Настоящая заявка испрашивает приоритет на основании предварительной заявки на патент США № 62/693766, поданной 3 июля 2018 года, полное содержание которой включено в настоящий документ посредством ссылки.
ОБЛАСТЬ ТЕХНИКИ
Настоящее изобретение относится к полифункциональным белковым молекулам, содержащим декорин, и их применениям. В частности, настоящее изобретение относится к полифункциональным белковым молекулам, содержащим декорин и нацеливающий полипептид, такой как антитело, а также способам их получения и их применениям.
УРОВЕНЬ ТЕХНИКИ
ФАКТОР РОСТА ЭНДОТЕЛИЯ СОСУДОВ (VEGF)
Ангиогенез представляет собой физиологический процесс, посредством которого новые кровеносные сосуды образуются из уже существующих сосудов. Он отличается от васкулогенеза, который представляет собой образование de novo эндотелиальных клеток из предшественников клеток мезодермы. Первые сосуды развивающегося эмбриона образуются посредством васкулогенеза, после чего ангиогенез ответственен за рост большинства, если не всех, кровеносных сосудов в процессе развития и при заболевании.
Ангиогенез представляет собой нормальный и жизненно важный процесс во время роста и развития, а также при заживлении ран и образовании грануляционной ткани. Однако он также является основным шагом в переходе опухолей из доброкачественного состояния в злокачественное, что обусловливает применение ингибиторов ангиогенеза для лечения рака.
Фактор роста эндотелия сосудов (VEGF), первоначально известный как фактор проницаемости сосудов (VPF), представляет собой сигнальный белок, вырабатываемый клетками, который стимулирует васкулогенез и ангиогенез. Он является частью системы, которая восстанавливает снабжение тканей кислородом при недостаточном кровообращении. Концентрация VEGF в сыворотке высока при бронхиальной астме и сахарном диабете. Нормальная функция VEGF заключается в создании новых кровеносных сосудов в процессе эмбрионального развития, новых кровеносных сосудов после травмы, мышц после физической нагрузки и новых сосудов (коллатеральное кровообращение) для обхода заблокированных сосудов.
Сверхэкспрессия VEGF может способствовать развитию заболевания. Солидные злокачественные новообразования не могут вырасти за пределы ограниченного размера без достаточного кровоснабжения; злокачественные новообразования, которые могут экспрессировать VEGF, способны к росту и метастазированию. Сверхэкспрессия VEGF может вызывать сосудистые заболевания сетчатки глаза и других частей организма. Лекарственные средства, такие как бевацизумаб и ранибизумаб, могут ингибировать VEGF и обеспечивать контроль или замедление развития этих заболеваний.
VEGF представляет собой подсемейство факторов роста, конкретнее, семейство тромбоцитарных факторов роста, относящихся к дисульфид-связанным факторам роста. Они являются важными сигнальными белками, участвующими как в васкулогенезе (образовании de novo эмбриональной системы кровообращения), так и в ангиогенезе (росте кровеносных сосудов из уже существующей сосудистой сети).
Ингибиторы контрольных точек
Важной частью иммунной системы является ее способность различать в организме нормальные клетки и клетки, которые она воспринимает как «чужеродные». Это позволяет иммунной системе атаковать чужеродные клетки, не затрагивая нормальные. Для этого иммунная система использует «контрольные точки» - молекулы на некоторых иммунных клетках, которые требуют активации (или инактивации) для запуска иммунного ответа. В некоторых случаях раковые клетки находят способы использования этих контрольных точек, позволяющие избежать атаки со стороны иммунной системы. Однако лекарственные средства, нацеленные на эти контрольные точки, представляются многообещающими в качестве средств для лечения рака.
Ингибиторы контрольных точек ориентированы на преодоление одной из основных защит раковых клеток против атаки со стороны иммунной системы. T-клетки иммунной системы постоянно «патрулируют» организм на присутствие признаков заболевания или инфекции. Встречаясь с другой клеткой, они проверяют наличие определенных белков на ее поверхности, которые служат признаками идентичности клетки. Если белки указывают на то, что клетка является нормальной и здоровой, T-клетка не затрагивает ее. Если белки свидетельствуют о том, что клетка является инфицированной или злокачественной, T-клетка будет ее атаковать. Как только T-клетки инициируют атаку, иммунная система увеличивает выработку ряда дополнительных молекул с целью предотвращения повреждения нормальных тканей организма вследствие атаки. Эти молекулы известны как иммунные контрольные точки.
Ингибиторы контрольных точек блокируют эти нормальные белки на раковых клетках или белки на T-клетках, которые восприимчивы к ним. В результате снимаются ограничения, которые мешали T-клеткам распознавать клетки как раковые и направлять атаку иммунной системы на них. Три ингибитора контрольных точек были быстро одобрены Федеральным управлением по контролю качества пищевых продуктов и лекарственных средств США (U.S.F.D.A.) для лечения рака, включая ипилимумаб (Ервой, Yervoy®), пембролизумаб (Кейтруда, Keytruda®) и ниволумаб (Опдиво, Opdivo®). Эти и другие лекарственные средства, нацеленные на иммунные контрольные точки, представляют одно из самых многообещающих направлений в лечении рака на сегодняшний день.
Существует потребность в дополнительных терапевтических агентах, которые нацелены на VEGF или иммунные контрольные точки.
КРАТКОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
Настоящее изобретение относится к полифункциональным белковым молекулам, содержащим декорин, и их применениям. В частности, настоящее изобретение относится к полифункциональным белковым молекулам, содержащим декорин и нацеливающий полипептид, такой как антитело, и способам их получения и их применениям.
Соответственно, в некоторых вариантах реализации настоящего изобретения предложены полифункциональные белковые молекулы, содержащие по меньшей мере одну молекулу декорина или ее функциональный участок, связанные с антигенсвязывающим белком.
В некоторых предпочтительных вариантах реализации антигенсвязывающий белок выбран из группы, состоящей из связывающего VEGF-A антигенсвязывающего белка и антигенсвязывающего белка ингибитора контрольной точки. В некоторых предпочтительных вариантах реализации антигенсвязывающий белок ингибитора контрольной точки связывает ингибирующий иммунный ответ белок контрольной точки, выбранный из группы, состоящей из PD-1, PD-L1, CTLA-4, PD-L2, CD27, CD28, CD40, CD47, CD115, CD122, CD137, OX40, GITR, ICOS, A2AR, B7-H3, B7-H4, BTLA, IDO, KIR, LAG3, NOX2, TIM-3, VISTA, SIGLEC-7, TIGIT и 4-1BB. В частности, в некоторых предпочтительных вариантах реализации антигенсвязывающий белок ингибитора контрольной точки связывает ингибирующий иммунный ответ белок контрольной точки, выбранный из группы, состоящей из PD-1, PD-L1, CTLA-4, PD-L2.
В некоторых предпочтительных вариантах реализации антигенсвязывающий белок представляет собой антитело. В некоторых предпочтительных вариантах реализации антитело представляет собой моноклональное антитело. В некоторых предпочтительных вариантах реализации моноклональное антитело выбрано из группы, состоящей из бевацизумаба, ранибизумаба, ипилимумаба, атезолизумаба, авелумаба, дурвалумаба, ниволумаба и пембролизумаба.
В некоторых предпочтительных вариантах реализации полипептид декорина представляет собой коровый белок декорина. В некоторых предпочтительных вариантах реализации коровый белок декорина содержит мутацию в положении 4 корового белка зрелого декорина. В некоторых предпочтительных вариантах реализации мутация представляет собой мутацию по типу замены серина на аланин. В некоторых предпочтительных вариантах реализации коровый белок декорина по существу не модифицирован молекулами гликозаминогликанов в положении 4 корового белка зрелого декорина. В некоторых предпочтительных вариантах реализации гибридный белок содержит две или более копий полипептида декорина.
В некоторых предпочтительных вариантах реализации по меньшей мере функциональный участок молекулы декорина содержит домен или домены декорина, которые связывают сигнальную молекулу, выбранную из группы, состоящей из трансформирующего фактора роста-β (TGF-β), фактора роста соединительной ткани (CTGF), тромбоцитарного фактора роста (PDGF), рецептора 2 фактора роста эндотелия сосудов (VEGFR2), рецептора фактора роста гепатоцитов (HGFR), рецептора инсулиноподобного фактора роста 1 (IGF-1R), рецепторов эпидермального фактора роста (EGFR), миостатина и C1q. В некоторых предпочтительных вариантах реализации связывающий TGF-β домен содержит аминокислоты Asp45-Lys359 полноразмерного эндогенного декорина человека или аминокислоты Leu155-Val260 полноразмерного эндогенного декорина человека. В некоторых предпочтительных вариантах реализации полифункциональная белковая молекула содержит две или более копий по меньшей мере функционального участка молекулы декорина.
В некоторых предпочтительных вариантах реализации молекула декорина функционально связана с тяжелой цепью антитела. В некоторых предпочтительных вариантах реализации антигенсвязывающий белок является биспецифичным. В некоторых предпочтительных вариантах реализации антигенсвязывающий белок является полиспецифичным.
В некоторых предпочтительных вариантах реализации полифункциональная белковая молекула представляет собой гибридный белок. В некоторых предпочтительных вариантах реализации молекула декорина химически связана с антигенсвязывающим белком.
В некоторых предпочтительных вариантах реализации настоящего изобретения предложены нуклеиновая кислота или совокупность нуклеиновых кислот, кодирующие полифункциональную белковую молекулу, описанную выше. В некоторых предпочтительных вариантах реализации настоящего изобретения предложены вектор или векторы, содержащие нуклеиновую кислоту или совокупность нуклеиновых кислот. В некоторых предпочтительных вариантах реализации настоящего изобретения предложена клетка-хозяин, содержащая указанные вектор или векторы.
В некоторых предпочтительных вариантах реализации настоящего изобретения предложены способы ингибирования белка-мишени и сигнальной молекулы в клетке, включающие: приведение клетки в контакт с полифункциональной белковой молекулой, молекулой нуклеиновой кислоты или вектором, описанными выше, при таких условиях, в которых по меньшей мере один вид активности белка-мишени и по меньшей мере один вид активности сигнального белка ингибированы в клетке, при этом белок-мишень выбран из группы, состоящей из VEGF-1, PD-1, PD-L1, CTLA-4, PD-L2, CD27, CD28, CD40, CD47, CD115, CD122, CD137, OX40, GITR, ICOS, A2AR, B7-H3, B7-H4, BTLA, IDO, KIR, LAG3, NOX2, TIM-3, VISTA, SIGLEC-7, TIGIT и 4-1BB. В некоторых предпочтительных вариантах реализации клетка представляет собой клетку in vitro или in vivo. В некоторых предпочтительных вариантах реализации клетка находится в субъекте. В некоторых предпочтительных вариантах реализации приведение в контакт приводит к ингибированию активности, выбранной из группы, состоящей из ангиогенеза, активности PD-1, активности PD-L1, активности CTLA-4, активности PD-L2, активности CD27, активности CD28, активности CD40, активности CD47, активности CD115, активности CD122, активности CD137, активности OX40, активности GITR, активности ICOS, активности A2AR, активности B7-H3, активности B7-H4, активности BTLA, активности IDO, активности KIR, активности LAG3, активности NOX2, активности TIM-3, активности VISTA, активности SIGLEC-7, активности TIGIT и активности 4-1BB. В некоторых предпочтительных вариантах реализации рак выбран из группы, состоящей из рака легкого, колоректального рака, рака печени, рака молочной железы, рака почки, рака шейки матки, рака яичника и глиобластомы. В некоторых предпочтительных вариантах реализации сигнальный белок выбран из группы, состоящей из трансформирующего фактора роста-β (TGF-β), фактора роста соединительной ткани (CTGF), тромбоцитарного фактора роста (PDGF), рецептора 2 фактора роста эндотелия сосудов (VEGFR2), рецептора фактора роста гепатоцитов (HGFR), рецептора инсулиноподобного фактора роста 1 (IGF-1R), различных рецепторов эпидермального фактора роста (EGFR), миостатина и C1q. В некоторых предпочтительных вариантах реализации сигнальный белок представляет собой трансформирующий фактор роста-β (TGF-β).
В некоторых предпочтительных вариантах реализации настоящего изобретения предложены способы лечения нарушения, характеризующегося ангиогенезом или ростом опухоли, включающие: введение субъекту полифункциональной белковой молекулы, молекулы нуклеиновой кислоты или вектора, описанных выше, при таких условиях, в которых ангиогенез или рост опухоли у субъекта подавляются. В некоторых предпочтительных вариантах реализации опухоль выбрана из группы, состоящей из рака легкого, колоректального рака, рака печени, рака молочной железы, рака почки, рака шейки матки, рака яичника и глиобластомы.
ОПИСАНИЕ ЧЕРТЕЖЕЙ
ФИГ. 1 представляет собой схематическое изображение гибридного белка согласно настоящему изобретению.
ФИГ. 2 представляет собой карту экспрессионной конструкции согласно настоящему изобретению.
ФИГ. 3 представляет собой карту экспрессионной конструкции согласно настоящему изобретению.
ФИГ. 4 представляет собой гель, полученный посредством электрофореза в полиакриламидном геле в присутствии додецилсульфата натрия (SDS-PAGE), для среды от объединенной популяции клеток линии CHO, экспрессирующих гибридный белок бевацизумаб. Дорожка 10. Стандарты молекулярной массы. Дорожка 11. Невосстановленный образец среды. Дорожка 12. Восстановленный образец среды.
ФИГ. 5 представляет собой гель, полученный посредством SDS-PAGE, для очищенной гибридной молекулы авелумаб-галакорин. Дорожка 1: маркеры молекулярной массы. Дорожка 2: невосстановленный очищенный гибридный белок авелумаб-галакорин. Дорожка 3: восстановленный очищенный гибридный белок авелумаб-галакорин.
ФИГ. 6 представляет собой хроматограмму, полученную посредством эксклюзионной ВЭЖХ, для очищенной гибридной молекулы авелумаб-галакорин. Процентное содержание мономера составляло более 98 %.
ФИГ. 7 представляет собой график роста опухоли в модели колоректального рака человека MC-38 у мышей C57BL/6 после внутривенного введения однократной дозы четырех препаратов.
ОПРЕДЕЛЕНИЯ
Для облегчения понимания настоящего изобретения ниже приведены определения ряда терминов.
В настоящем документе термин «декорин» относится к белковой молекуле, имеющей последовательность зрелого белка, которая на по меньшей мере 80 % идентична SEQ ID NO: 1, 2 или 6 или их части.
В настоящем документе термин «коровый белок декорина» относится к молекуле белка декорина, которая содержит мутацию в положении аминокислоты 4 зрелого декорина и по существу не модифицирована гликозаминогликаном (ГАГ, то есть не гагилирована) по аминокислоте 4.
В настоящем документе термин «полифункциональная белковая молекула» относится к белковым молекулам, которые содержат две или более полипептидных подчастей, полученных по меньшей мере из двух разных источников. Полифункциональные белковые молекулы могут представлять собой рекомбинантные гибридные белки, кодируемые гибридным геном, или могут быть получены путем химического присоединения (например, путем ковалентной модификации) полипептида к другому полипептиду. Например, гибридные белки согласно настоящему изобретению могут предпочтительно содержать одну или более молекул декорина или их функциональных участков, связанных с антигенсвязывающим белком посредством линкерной последовательности, либо могут представлять собой «химические гибридные белки», в которых одна или более молекул декорина или их функциональных участков ковалентно связаны с антигенсвязывающим белком, например, посредством модифицированной аминокислоты.
В настоящем документе термин «клетка-хозяин» относится к любой эукариотической клетке (например, клеткам млекопитающих, клеткам птиц, клеткам земноводных, клеткам растений, клеткам рыб и клеткам насекомых) независимо от того, находится ли клетка в условиях in vitro или in vivo.
В настоящем документе термин «клеточная культура» относится к любой культуре клеток in vitro. Этот термин включает стабильные клеточные линии (например, имеющие иммортализованный фенотип), первичные клеточные культуры, перевиваемые клеточные линии (например, нетрансформированные клетки) и любую другую популяцию клеток, поддерживаемую in vitro, включая ооциты и эмбрионы.
В настоящем документе термин «вектор» относится к любому генетическому элементу, такому как плазмида, фаг, транспозон, космида, хромосома, вирус, вирион и так далее, который способен к репликации, когда он связан с соответствующими регуляторными элементами, и который может переносить последовательности генов между клетками. Таким образом, термин включает переносчики для клонирования и экспрессии, а также вирусные векторы.
В настоящем документе термины «комплементарный» или «комплементарность» используются в отношении полинуклеотидов (то есть последовательности нуклеотидов), связанных в соответствии с правилами спаривания оснований. Например, последовательность «5'-A-G-T-3'» комплементарна последовательности «3'-T-C-A-5'». Комплементарность может быть «частичной», при которой только некоторые из оснований нуклеиновых кислот соответствуют правилам спаривания оснований. Либо, комплементарность между нуклеиновыми кислотами может быть «полной» или «абсолютной». Степень комплементарности между цепями нуклеиновых кислот оказывает значительное влияние на эффективность и силу гибридизации между цепями нуклеиновых кислот. Это особенно важно в реакциях амплификации, а также в методах детекции, зависящих от связывания нуклеиновых кислот.
Термины «гомология» и «процент идентичности», используемые в отношении нуклеиновых кислот, относятся к степени комплементарности. Гомология может быть частичной (то есть частичная идентичность) или полной (то есть полная идентичность). Частично комплементарная последовательность представляет собой последовательность, которая по меньшей мере частично ингибирует гибридизацию полностью комплементарной последовательности с последовательностью нуклеиновой кислоты-мишени, и ее обозначают с помощью функционального термина «по существу гомологичная». Ингибирование гибридизации полностью комплементарной последовательности к последовательностью-мишенью может быть исследовано с применением анализа гибридизации (Саузерн-блоттинг или Нозерн-блоттинг, гибридизация в растворе и тому подобное) в условиях низкой жесткости. По существу гомологичные последовательность или зонд (то есть олигонуклеотид, который способен гибридизоваться с другим целевым олигонуклеотидом) будут конкурировать за последовательность-мишень и ингибировать ее связывание (то есть гибридизацию) с полностью гомологичной последовательностью с в условиях низкой жесткости. Это не означает, что условия низкой жесткости допускают неспецифичное связывание; условия низкой жесткости требуют, чтобы связывание двух последовательностей друг с другом являлось специфичным (то есть селективным) взаимодействием. Отсутствие неспецифичного связывания может быть проверено путем применения второй мишени, у которой отсутствует даже частичная степень комплементарности (например, имеющей идентичность менее примерно 30 %); в отсутствие неспецифичного связывания зонд не будет гибридизоваться со второй некомплементарной мишенью.
Термины «в функциональной комбинации», «в функциональном порядке» и «функционально связанный» в настоящем документе относятся к такой связи между последовательностями нуклеиновых кислот, при которой молекула нуклеиновой кислоты способна управлять транскрипцией данного гена и/или обеспечивает синтез желаемой белковой молекулы. Указанный термин также относится к такой связи между аминокислотными последовательностями, при которой образуется функциональный белок.
В настоящем документе термин «сигнальная последовательность» относится к любой последовательности ДНК, которая, являясь функционально связанной с последовательностью рекомбинантной ДНК, кодирует сигнальный пептид, способный вызывать секрецию рекомбинантного полипептида. Обычно сигнальные пептиды содержат последовательность из примерно 15-30 гидрофобных аминокислотных остатков (см., например, Zwizinski et al., J. Biol. Chem. 255(16): 7973-77 [1980], Gray et al., Gene 39(2): 247-54 [1985] и Martial et al., Science 205: 602-607 [1979]). Такие секреторные сигнальные последовательности предпочтительно получены из генов, которые кодируют полипептиды, секретируемые из типов клеток, нацеленных на тканеспецифичную экспрессию (например, секретируемые белки молока для экспрессии в и секреции из секреторных клеток молочной железы). Однако секреторные последовательности ДНК не ограничены такими последовательностями. Также могут применяться секреторные последовательности ДНК для белков, секретируемых из многих типов клеток и организмов (например, сигналы секреции тканевого активатора плазминогена (t-PA), сывороточного альбумина, лактоферрина и гормона роста и сигналы секреции из генов микроорганизмов, кодирующих секретируемые полипептиды, например, из генов дрожжей, мицелиальных грибов и бактерий).
В настоящем документе термин «очищенный» относится к молекулам, последовательностям нуклеиновых кислот или аминокислот, которые извлечены из их природной среды, выделены или разделены. Таким образом, «последовательность выделенной нуклеиновой кислоты» представляет собой последовательность очищенной нуклеиновой кислоты. «По существу очищенные» молекулы на по меньшей мере 60 %, предпочтительно на по меньшей мере 75 % и более предпочтительно на по меньшей мере 90 % не содержат других компонентов, с которыми они обычно связаны.
«Акцепторная каркасная область человека» для целей настоящего изобретения представляет собой каркасную область, содержащую аминокислотную последовательность каркасной области вариабельного домена легкой цепи (VL) или каркасной области вариабельного домена тяжелой цепи (VH), полученной из каркасной области иммуноглобулина человека или консенсусной каркасной области человека, определенных ниже. Акцепторная каркасная область человека, «полученная из» каркасной области иммуноглобулина человека или консенсусной каркасной области человека, может содержать ту же самую аминокислотную последовательность, либо она может содержать измененную аминокислотную последовательность. В некоторых вариантах реализации количество замен аминокислот составляет 10 или меньше, 9 или меньше, 8 или меньше, 7 или меньше, 6 или меньше, 5 или меньше, 4 или меньше, 3 или меньше или 2 или меньше. В некоторых вариантах реализации последовательность акцепторной каркасной области VL человека идентична последовательности каркасной области VL иммуноглобулина человека или последовательности консенсусной каркасной области человека.
«Аффинность» относится к силе совокупных нековалентных взаимодействий между отдельным сайтом связывания молекулы (например, антитела) и ее партнером по связыванию (например, антигеном). Если не указано иное, в настоящем документе «аффинность связывания» относится к истинной аффинности связывания, которая отражает взаимодействие 1:1 между членами пары связывания (например, антителом и антигеном). Аффинность молекулы X для ее партнера Y может в общем быть представлена с помощью константы диссоциации (Kd). Аффинность может быть определена обычными способами, известными в данной области техники, включая способы, описанные в настоящем документе. Конкретные иллюстративные и типовые варианты реализации для определения аффинности связывания описаны ниже.
«Антитело с созревшей аффинностью» относится к антителу с одной или более модификациями в одной или более гипервариабельных областях (HVR) по сравнению с исходным антителом, которое не имеет таких модификаций, приводящих к повышению аффинности антитела к антигену.
Термин «антитело» в настоящем документе используется в самом широком смысле и охватывает различные структуры антител, включая, но не ограничиваясь ими, моноклональные антитела, поликлональные антитела, полиспецифичные антитела (например, биспецифичные антитела) и фрагменты антител, если они проявляют желаемую антигенсвязывающую активность.
«Фрагмент антитела» относится к молекуле, отличной от интактного антитела, содержащей участок интактного антитела и связывающей антиген, с которым связывается интактное антитело. Примеры фрагментов антител включают, но не ограничиваются указанными, Fv, Fab, Fab', Fab'-SH, F(ab')2; диатела; линейные антитела; одноцепочечные молекулы антител (например, scFv); и полиспецифичные антитела, образованные из фрагментов антител.
«Антитело, которое связывается с тем же эпитопом», что и эталонное антитело, относится к антителу, которое на 50 % или больше блокирует связывание эталонного антитела с его антигеном в конкурентном анализе, и, наоборот, эталонное антитело на 50 % или больше блокирует связывание указанного антитела с его антигеном в конкурентном анализе. В настоящем документе предложен типичный конкурентный анализ.
Термин «химерное антитело» относится к антителу, в котором участок тяжелой и/или легкой цепи получен из конкретного источника или вида, тогда как остальная часть тяжелой и/или легкой цепи получена из другого источника или вида.
«Класс» антитела относится к типу константного домена или константной области, содержащихся в тяжелой цепи антитела. Существуют пять основных классов антител: IgA, IgD, IgE, IgG и IgM, и некоторые из них могут быть дополнительно разделены на подклассы (изотипы), например, IgG1, IgG2, IgG3, IgG4, IgA1 и IgA2. Константные домены тяжелой цепи, которые соответствуют различным классам иммуноглобулинов, называют α, δ, ε, γ и μ, соответственно.
«Эффекторные функции» относятся к видам биологической активности, присущей Fc-области антитела, которая варьирует в зависимости от изотипа антитела. Примеры эффекторных функций антитела включают: связывание C1q и комплементзависимую цитотоксичность (КЗЦ); связывание с Fc-рецептором; антителозависимую клеточно-опосредованную цитотоксичность (АЗКЦ); фагоцитоз; понижающую регуляцию рецепторов клеточной поверхности (например, B-клеточного рецептора); и активацию B-клеток.
«Эффективное количество» агента, например, фармацевтического состава, относится к количеству, являющемуся эффективным в дозировках и в течение периодов времени, необходимых для достижения желаемого терапевтического или профилактического результата.
Термин «эпитоп» относится к конкретному сайту на молекуле антигена, с которым связывается антитело.
Термин «Fc-область» в настоящем документе используется для определения C-концевой области тяжелой цепи иммуноглобулина, которая содержит по меньшей мере часть константной области. Термин включает Fc-области, имеющие природную последовательность, и вариантные Fc-области. В одном варианте реализации Fc-область тяжелой цепи IgG человека простирается от Cys226 или от Pro230 до C-конца тяжелой цепи. Однако C-концевой лизин (Lys447) Fc-области может присутствовать или отсутствовать. Если в настоящем документе не указано иное, нумерация аминокислотных остатков в Fc-области или константной области соответствует системе нумерации EU, также называемой индексом EU, описанной в Kabat et al., Sequences of Proteins of Immunological Interest, 5th Ed. Public Health Service, National Institutes of Health, Bethesda, MD, 1991.
«Каркасная область» или «FR» относится к остаткам вариабельного домена, отличным от остатков гипервариабельной области (HVR). FR вариабельного домена обычно состоит из четырех доменов FR: FR1, FR2, FR3 и FR4. Соответственно, последовательности HVR и FR обычно появляются в следующей последовательности в VH (или VL): FR1-H1(L1)-FR2-H2(L2)-FR3-H3(L3)-FR4.
Термины «полноразмерное антитело», «интактное антитело» и «целое антитело» в настоящем документе используются взаимозаменяемо для обозначения антитела, имеющего структуру, по существу схожую со структурой природного антитела, или содержащего тяжелые цепи, которые содержат Fc-область, определенную в настоящем документе.
Термины «клетка-хозяин», «линия клеток-хозяев» и «культура клеток-хозяев» используются взаимозаменяемо и относятся к клеткам, в которые была введена экзогенная нуклеиновая кислота, включая потомство таких клеток. Клетки-хозяева включают «трансформанты» и «трансформированные клетки», которые включают первичную трансформированную клетку и полученное от нее потомство независимо от количества пересевов. Потомство может не быть полностью идентичным родительской клетке по содержанию нуклеиновых кислот, при этом может содержать мутации. Настоящее изобретение включает мутантное потомство, которое имеет те же самые функцию или биологическую активность, что и подвергнутые скринингу или отобранные в первоначально трансформированной клетке.
«Антитело человека» представляет собой антитело, которое имеет аминокислотную последовательность, соответствующую последовательности антитела, вырабатываемого человеком или клеткой человека, или полученного из отличного от человека источника, которое использует спектр антител человека или другие последовательности, кодирующие антитела человека. Это определение антитела человека конкретно исключает гуманизированное антитело, содержащее антигенсвязывающие остатки, отличные от антигенсвязывающих остатков антитела человека.
«Консенсусная каркасная область человека» представляет собой каркасную область, которая представляет наиболее часто встречающиеся аминокислотные остатки в совокупности последовательностей каркасной области VL или VH иммуноглобулина человека. Как правило, последовательности VL или VH иммуноглобулина человека выбраны из подгруппы последовательностей вариабельных доменов. Обычно указанная подгруппа последовательностей представляет собой подгруппу, описанную в источнике Kabat et al., Sequences of Proteins of Immunological Interest, Fifth Edition, NIH Publication 91-3242, Bethesda MD (1991), vols. 1-3. В одном варианте реализации, для VL, подгруппа представляет собой подгруппу каппа I, описанную в источнике Kabat et al., см. выше. В одном варианте реализации, для VH, подгруппа представляет собой подгруппу III, описанную в источнике Kabat et al., см. выше.
«Гуманизированное» антитело относится к химерному антителу, содержащему аминокислотные остатки из HVR, отличных от HVR человека, и аминокислотные остатки из FR человека. В некоторых вариантах реализации гуманизированное антитело будет содержать по существу все из по меньшей мере одного, а обычно двух, вариабельных доменов, в которых все или по существу все HVR (например, CDR) соответствуют таковым для антитела, отличного от антитела человека, и все или по существу все FR соответствуют таковым для антитела человека. Гуманизированное антитело необязательно может содержать по меньшей мере часть константной области антитела, полученной из антитела человека. «Гуманизированная форма» антитела, например, антитела, отличного от антитела человека, относится к антителу, которое подвергли гуманизации было гуманизировано.
Термин «гипервариабельная область» или «HVR» в настоящем документе относится к каждой из областей вариабельного домена антитела, которые являются гипервариабельными по последовательности и/или образуют структурно определенные петли («гипервариабельные петли»). Как правило, природные четырехцепочечные антитела содержат шесть HVR; три в VH (H1, H2, H3) и три в VL (L1, L2, L3). HVR обычно содержат аминокислотные остатки из гипервариабельных петель и/или из «областей, определяющих комплементарность» (CDR), причем последние характеризуются наибольшей вариабельностью последовательностей и/или участвуют в распознавании антигена. Типичные гипервариабельные петли встречаются в положениях аминокислотных остатков 26-32 (L1), 50-52 (L2), 91-96 (L3), 26-32 (H1), 53-55 (H2) и 96-101 (H3). (Chothia and Lesk, J. Mol. Biol. 196:901-917 (1987).) Типичные CDR (CDR-L1, CDR-L2, CDR-L3, CDR-H1, CDR-H2 и CDR-H3) встречаются в положениях аминокислотных остатков 24-34 петли L1, 50-56 петли L2, 89-97 петли L3, 31-35B петли H1, 50-65 петли H2 и 95-102 петли H3. (Kabat et al., Sequences of Proteins of Immunological Interest, 5th Ed. Public Health Service, National Institutes of Health, Bethesda, MD (1991).) За исключением CDR1 в VH, обычно CDR содержат аминокислотные остатки, которые образуют гипервариабельные петли. CDR также содержат «остатки, определяющие специфичность», или «SDR», которые представляют собой остатки, вступающие в контакт с антигеном. SDR содержатся в областях CDR, называемых «укороченные CDR» (abbreviated-CDR), или a-CDR. Типичные a-CDR (a-CDR-L1, a-CDR-L2, a-CDR-L3, a-CDR-H1, a-CDR-H2 и a-CDR-H3) встречаются в положениях аминокислотных остатков 31-34 петли L1, 50-55 петли L2, 89-96 петли L3, 31-35B петли H1, 50-58 петли H2 и 95-102 петли H3. (См. Almagro and Fransson, Front. Biosci. 13:1619-1633 (2008).) Если не указано иное, остатки HVR и другие остатки в вариабельном домене (например, остатки FR) пронумерованы в настоящем документе в соответствии с источником Kabat et al., см. выше.
«Индивидуум» или «субъект» представляет собой млекопитающее. Млекопитающие включают, но не ограничиваются указанными, домашних животных (например, коров, овец, кошек, собак и лошадей), приматов (например, людей и отличных от человека приматов, таких как обезьяны), кроликов и грызунов (например, мышей и крыс). В некоторых вариантах реализации индивидуум или субъект представляет собой человека.
«Выделенное антитело» представляет собой антитело, которое было отделено от компонента его природного окружения. В некоторых вариантах реализации антитело очищают до степени чистоты более 95 % или 99 %, определяемой, например, посредством электрофоретического анализа (например, SDS-PAGE, изоэлектрического фокусирования (ИЭФ), капиллярного электрофореза) или хроматографического анализа (например, ионообменной или обращенно-фазовой ВЭЖХ). Обзор методов оценки чистоты антител см., например, в источнике Flatman et al., J. Chromatogr. B 848:79-87 (2007).
«Выделенная нуклеиновая кислота» относится к молекуле нуклеиновой кислоты, которая была отделена от компонента ее природного окружения. Выделенная нуклеиновая кислота включает молекулу нуклеиновой кислоты, содержащуюся в клетках, которые обычно содержат молекулу нуклеиновой кислоты, но указанная молекула нуклеиновой кислоты находится вне хромосом или имеет хромосомную локализацию, отличную от ее природной хромосомной локализации.
Термин «моноклональное антитело» в настоящем документе относится к антителу, полученному из популяции по существу гомогенных антител, то есть отдельные антитела, составляющие популяцию, являются идентичными и/или связывают один и тот же эпитоп, за исключением возможных вариантных антител, например, содержащих природные мутации или возникающие в процессе получения препарата моноклонального антитела, при этом такие варианты обычно присутствуют в незначительных количествах. В отличие от препаратов поликлональных антител, которые обычно содержат разные антитела, направленные против разных детерминант (эпитопов), каждое моноклональное антитело в препарате моноклонального антитела направлено против единственной детерминанты на молекуле антигена. Таким образом, определение «моноклональное» указывает на характер антитела как полученного из по существу гомогенной популяции антител, и оно не подразумевает получение антитела с применением какого-либо конкретного способа. Например, моноклональные антитела для применения в соответствии с настоящим изобретением могут быть получены с помощью различных методик, включая, но не ограничиваясь ими, гибридомный метод, методы рекомбинантной ДНК, методы фагового дисплея и методы с использованием трансгенных животных, содержащих все локусы иммуноглобулина человека или их часть; такие способы и другие иллюстративные способы получения моноклональных антител описаны в настоящем документе.
«Природные антитела» относятся к природным молекулам иммуноглобулинов с различными структурами. Например, природные антитела IgG представляют собой гетеротетрамерные гликопротеины с молекулярной массой примерно 150000 дальтон, состоящие из двух идентичных легких цепей и двух идентичных тяжелых цепей, связанных посредством дисульфидной связи. От N- до C-конца каждая тяжелая цепь содержит вариабельную область (VH), также называемую вариабельным тяжелым доменом или вариабельным доменом тяжелой цепи, за которой следуют три константных домена (CH1, CH2 и CH3). Аналогичным образом, от N- до C-конца каждая легкая цепь содержит вариабельную область (VL), также называемую вариабельным легким доменом или вариабельным доменом легкой цепи, за которой следует константный домен легкой цепи (CL). Легкая цепь антитела может быть отнесена на основе аминокислотной последовательности ее константного домена к одному из двух типов, называемых каппа (κ) и лямбда (λ).
«Процент (%) идентичности аминокислотной последовательности» по отношению к эталонной полипептидной последовательности определяют как процент аминокислотных остатков в последовательности-кандидате, которые идентичны аминокислотным остаткам в эталонной полипептидной последовательности после выравнивания последовательностей и внесения пропусков, при необходимости, для достижения максимального процента идентичности последовательностей, причем, без учета каких-либо консервативных замен в качестве составляющих идентичности последовательностей. Выравнивание с целью определения процента идентичности аминокислотных последовательностей может быть осуществлено различными способами, известными специалистам в данной области техники, например, с использованием находящегося в открытом доступе компьютерного программного обеспечения, такого как программное обеспечение BLAST, BLAST-2, ALIGN или Megalign (DNASTAR). Специалисты в данной области техники могут определить подходящие параметры для выравнивания последовательностей, включая любые алгоритмы, необходимые для достижения максимального выравнивания в пределах всей длины сравниваемых последовательностей. Однако для целей настоящего изобретения значения % идентичности аминокислотных последовательностей получают с использованием компьютерной программы для сравнения последовательностей ALIGN-2. Компьютерная программа для сравнения последовательностей ALIGN-2 была разработана Genentech, Inc., а исходный код был подан вместе с пользовательской документацией в Бюро регистрации авторских прав США, Вашингтон, округ Колумбия, 20559, где он зарегистрирован под регистрационным номером авторских прав США TXU510087. Программа ALIGN-2 находится в открытом доступе от Genentech, Inc., Южный Сан-Франциско, Калифорния, либо может быть скомпилирована из исходного кода. Программа ALIGN-2 должна быть скомпилирована для использования в операционной системе UNIX, включая Digital UNIX V4.0D. Все параметры сравнения последовательностей заданы программой ALIGN-2 и не меняются.
В тех ситуациях, когда ALIGN-2 используют для сравнения аминокислотных последовательностей, % идентичности аминокислотных последовательностей данной аминокислотной последовательности A с, вместе с или против данной аминокислотной последовательности B (что, в качестве альтернативы, может быть сформулировано как данная аминокислотная последовательность A, которая имеет или содержит определенный % идентичности аминокислотных последовательностей с, вместе с или против данной аминокислотной последовательности B) рассчитывают следующим образом:
Отношение X/Y, умноженное на 100,
где X представляет собой число аминокислотных остатков, оцененных программой для выравнивания последовательностей ALIGN-2 как идентичные совпадения при выравнивании A и B в этой программе, и где Y представляет собой общее число аминокислотных остатков в B. Следует понимать, что в случае, когда длина аминокислотной последовательности A не равна длине аминокислотной последовательности B, % идентичности аминокислотных последовательностей при сравнении A с B не будет равен % идентичности аминокислотных последовательностей при сравнении B с A. Если конкретно не указано иное, все используемые в настоящем документе значения % идентичности аминокислотных последовательностей получены, как описано в предыдущем абзаце, с помощью компьютерной программы ALIGN-2.
Термин «фармацевтический состав» относится к препарату, который находится в форме, обеспечивающей эффективное проявление биологической активности содержащегося в нем активного ингредиента, и который не содержит дополнительных компонентов, обладающих неприемлемой токсичностью для субъекта, которому будет введен состав.
«Фармацевтически приемлемый носитель» относится к ингредиенту фармацевтического состава, отличному от активного ингредиента, который является нетоксичным для субъекта. Фармацевтически приемлемый носитель включает, но не ограничивается ими, буфер, вспомогательное вещество, стабилизатор или консервант.
В настоящем документе термин «лечение» (и его грамматические вариации, такие как «лечить» или «лечащий») относится к клиническому вмешательству в попытке изменить естественное развитие заболевания у подлежащего лечению индивидуума, которое может быть проведено либо в целях профилактики, либо во время развития клинической патологии. Желаемые эффекты лечения включают, но не ограничиваются указанными, предотвращение возникновения или рецидива заболевания, облегчение симптомов, уменьшение любых непосредственных или опосредованных патологических последствий заболевания, предотвращение метастазирования, уменьшение темпа прогрессирования заболевания, улучшение или временное облегчение патологического состояния и ремиссию или улучшенный прогноз. В некоторых вариантах реализации антитела согласно настоящему изобретению применяют для задерживания развития заболевания или для замедления прогрессирования заболевания.
Термин «вариабельная область» или «вариабельный домен» относится к домену тяжелой или легкой цепи антитела, который участвует в связывании антитела с антигеном. Вариабельные домены тяжелой цепи и легкой цепи (VH и VL, соответственно) природного антитела обычно имеют схожие структуры, причем каждый домен содержит четыре консервативных каркасных области (FR) и три гипервариабельные области (HVR). (См., например, Kindt et al. Kuby Immunology, 6th ed., W.H. Freeman and Co., page 91 (2007).) Присутствие одного домена VH или VL может быть достаточно для придания специфичности связывания с антигеном. Кроме того, антитела, которые связывают конкретный антиген, могут быть выделены с помощью домена VH или VL из антитела, которое связывает антиген, для скрининга библиотеки комплементарных доменов VL или VH, соответственно. См., например, Portolano et al., J. Immunol. 150:880-887 (1993); Clarkson et al., Nature 352:624-628 (1991).
В настоящем документе термин «вектор» относится к молекуле нуклеиновой кислоты, способной обеспечивать репродукцию другой нуклеиновой кислоты, с которой она связана. Указанный термин включает вектор как самореплицирующуюся нуклеотидную структуру, а также вектор, встроенный в геном клетки-хозяина, в которую он был введен. Некоторые векторы способны управлять экспрессией нуклеиновых кислот, с которыми они функционально связаны. В настоящем документе такие векторы обозначены термином «векторы экспрессии».
ПОДРОБНОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
Настоящее изобретение относится к полифункциональным молекулам, содержащим молекулу декорина или ее функциональный участок, которые функционально связаны с антигенсвязывающим белком, таким как молекула иммуноглобулина, укороченный фрагмент молекулы антигена или одноцепочечное антитело. В частности, настоящее изобретение относится к гибридным полипептидам, содержащим декорин и полипептид, нацеленный на VEGF или иммунную контрольную точку (предпочтительно ингибитор контрольной точки), и к способам их получения и их применениям. Варианты реализации настоящего изобретения обеспечивают гибридные полипептиды, содержащие полипептид декорина, слитый с целевым антигенсвязывающим белком, нуклеиновые кислоты, кодирующие такие полипептиды, и их применения. В настоящем документе описаны иллюстративные композиции и способы. Декорин, применяемый в гибридных молекулах, может представлять собой декорин дикого типа, коровый белок декорина или функциональные участки любого из этих белков, такие как участок(и), связывающий(ие) TGF-β или другие сигнальные молекулы, такие как фактор роста соединительной ткани (CTGF), тромбоцитарный фактор роста (PDGF), рецептор 2 фактора роста эндотелия сосудов (VEGFR2), рецептор фактора роста гепатоцитов (HGFR), рецептор инсулиноподобного фактора роста 1 (IGF-1R), различные рецепторы эпидермального фактора роста (EGFR), миостатин и C1q.
I. Декорин
В предпочтительных вариантах реализации полифункциональные белковые молекулы согласно настоящему изобретению содержат один или более полипептидов декорина или их функциональных участков. Было показано, что декорин подавляет индуцируемую трансформирующим фактором роста-бета экспрессию ингибитора активатора плазминогена-1 (см., например, источник Wahab et al., Biochem J. 2002 Mar 15; 362(Pt 3): 643-649; который полностью включен в настоящий документ посредством ссылки). Трансформирующий фактор роста-бета (TGF-β) является ключевым посредником накопления внеклеточного матрикса (ВКМ) при склеротических заболеваниях почек, таких как диабетическая нефропатия. Без ограничения конкретным механизмом, предполагается, что комбинация декорина и связывающего VEGF белка будет улучшать эффективность по сравнению со связывающим VEGF белком отдельно за счет ингибирования активности TGF-β в сочетании с блокированием VEGF.
Природный декорин представляет собой гликопротеин, содержащий присоединенный гликозаминогликан и имеющий среднюю молекулярную массу 90-140 кДа. В некоторых предпочтительных вариантах реализации декорин представляет собой коровый белок декорина, то есть по существу негагилированный декорин. В некоторых вариантах реализации коровый белок декорина содержит мутацию в положении аминокислоты 4 (то есть 4ой аминокислоты от N-конца) молекулы корового белка зрелого декорина. В некоторых вариантах реализации мутация представляет собой мутацию по типу замены серина на аланин. В некоторых вариантах реализации коровый белок декорина на по меньшей мере 90 %, 95 %, 99 % или 100 % идентичен SEQ ID NO:6 (коровый белок зрелого декорина) с учетом того, что коровый белок декорина содержит мутацию в положении аминокислоты 4 (то есть 4ой аминокислоты от N-конца) молекулы корового белка зрелого декорина.
Декорин обычно экспрессируется в виде препробелка. В настоящем изобретении предложены полифункциональные белковые молекулы, содержащие антигенсвязывающий белок, функционально связанный с одной или более последовательностями зрелого пептида декорина или его функциональных участков. В некоторых вариантах реализации часть корового белка декорина гибридного полипептида на по меньшей мере 90 %, 95 %, 99 % или 100 % идентична SEQ ID NO:6 (коровый белок зрелого декорина) или его функционального участка. В некоторых вариантах реализации коровый белок декорина содержит мутацию в положении аминокислоты 4 (то есть 4ой аминокислоты от N-конца) молекулы корового белка зрелого декорина.
В настоящем изобретении дополнительно предложены нуклеотидные последовательности, кодирующие гибридные белки, а также векторы, содержащие указанные нуклеотидные последовательности. В некоторых вариантах реализации часть корового белка декорина гибридного полипептида на по меньшей мере 90 %, 95 %, 99 % или 100 % идентична SEQ ID NO:5 (коровый белок зрелого декорина) с учетом того, что коровый белок декорина содержит мутацию в положении аминокислоты 4 (т. е. 4ой аминокислоты от N-конца) молекулы корового белка зрелого декорина.
В некоторых вариантах реализации молекула декорина, применяемая в полифункциональных белковых молекулах, может содержать один или более функциональных участков молекулы декорина. Молекулы декорина содержат ряд функциональных участков или доменов, например, описанных в источнике Järvinen and Prince, BioMed Research Int’l,Vol. 2015, Article ID 654765 (который полностью включен в настоящий документ посредством ссылки). В некоторых предпочтительных вариантах реализации функциональный участок декорина связывается с или иным образом взаимодействует с трансформирующим фактором роста-β (TGF-β), фактором роста соединительной ткани (CTGF), тромбоцитарным фактором роста (PDGF), рецептором 2 фактора роста эндотелия сосудов (VEGFR2), рецептором фактора роста гепатоцитов (HGFR), рецептором инсулиноподобного фактора роста 1 (IGF-1R), различными рецепторами эпидермального фактора роста (EGFR), миостатином или C1q. Предпочтительные функциональные участки декорина сохраняют функциональную активность (такую как связывание с одной из описанных непосредственно выше сигнальных молекул) и предпочтительно на по меньшей мере 90 %, 95 %, 99 % или 100 % идентичны соответствующей последовательности природного декорина. Предпочтительные функциональные участки декорина более короткие, чем полноразмерная молекула природного декорина, и, например, могут иметь длину от 10 до 300 аминокислот или от 10 до 120 аминокислот. Например, функциональный участок молекулы декорина может быть на по меньшей мере 90 %, 95 %, 99 % или 100 % идентичным SEQ ID NO:64 (связывающий TGF-β домен декорина, ASP45-LYS359 полноразмерного эндогенного декорина человека), SEQ ID NO:65 (две копии связывающего TGF-β домена декорина, ASP45-LYS359, разделенные линкером), SEQ ID NO:66 (связывающий TGF-β домен декорина, LEU155-VAL260 полноразмерного эндогенного декорина человека), SEQ ID NO:67 (две копии связывающего TGF-β домена декорина, LEU155-VAL260, разделенные линкером).
Иллюстративные полипептиды декорина и способ очистки декорина описаны, например, в источнике WO 2006038107, который полностью включен в настоящий документ посредством ссылки. Иллюстративные нуклеотидные и аминокислотные последовательности декорина представлены ниже и на фигурах 1 и 2.
Коровый белок декорина
Пропептид декорина
Пропептид декорина
Коровый белок декорина
Связывающий TGF-β домен декорина ASP45-LYS359
Связывающий TGF-β домен декорина ASP45-LYS359, две копии, разделенные линкером
Связывающий TGF-β домен декорина LEU155-VAL260
Связывающий TGF-β домен декорина LEU155-VAL260
Две копии, разделенные линкером
II. Связывающие агенты
В предпочтительных вариантах реализации настоящего изобретения предложены полифункциональные белковые молекулы, содержащие одну или более молекул декорина или ее функционального (функциональных) участка (участков), которые функционально связаны с целевым связывающим агентом. Предпочтительные целевые связывающие агенты включают, но не ограничиваются указанными, антигенсвязывающие белки, включая иммуноглобулины и их фрагменты или производные, такие как одноцепочечные антитела, которые связываются с такими молекулами как VEGF (фактор роста эндотелия сосудов) и ингибирующими иммунный ответ белками контрольных точек, такими как CTLA-4 (ассоциированный с цитотоксическими T-лимфоцитами антиген 4), PD-1 (белок 1 программируемой гибели клеток), PD-L1 (лиганд 1 белка программируемой гибели клеток), PD-L2 (лиганд 2 белка программируемой гибели клеток), CD27, CD28, CD40, CD47, CD115, CD122, CD137, OX40, GITR, ICOS, A2AR, B7-H3, B7-H4, BTLA, IDO, KIR, LAG3, NOX2, TIM-3, VISTA, SIGLEC-7, TIGIT и 4-1BB.
Связывающие агенты (например, антигенсвязывающие белки) обычно взаимодействуют c или специфично связываются с мишенью. Например, раскрытые в настоящем документе связывающие агенты обычно специфично взаимодействуют с областями, например, VEGF, CTLA-4, PD-1 или PD-L1, которые в настоящем документе в совокупности называют белками-мишенями. «Специфичное» связывание с белком-мишенью означает, что степень связывания с белком-мишенью больше, чем степень связывания с белками, отличными от белка-мишени (например, может наблюдаться фоновое неспецифичное связывание). Как правило, специфичное связывание связывающих агентов с белком, например, может быть достигнуто путем связывания со специфической последовательностью аминокислот в белке-мишени. Эти последовательности могут быть названы эпитопами. Молекулы, содержащие эпитопы, могут применяться для стимулирования связывающих агентов, таких как антитела, и могут быть названы иммуногенами. Связывающие агенты также могут распознавать специфические 2- и/или 3-мерные структуры как часть эпитопа. Антигенсвязывающий белок может быть моноспецифичным, биспецифичным или полиспецифичным.
Специфичное взаимодействие или связывание связывающего агента с его мишенью относят к типу равновесной реакции. В одном примере специфичное связывание может быть определено количественно. Для количественного определения может быть использована константа диссоциации, или Kd, известная в данной области техники как тип константы равновесия, которая описывает склонность, в данном случае антитела, отделяться от антигена или эпитопа, с которыми оно связано. Таким образом, Kd описывает аффинность антитела к эпитопу. Чем меньше Kd, тем больше аффинность связывающего агента к его мишени.
В некоторых вариантах реализации связывающий агент представляет собой моноклональное антитело. Антитело (например, моноклональное антитело) также может относиться к любому подходящему изотипу или подклассу изотипа. Связывающий агент также может представлять собой производное антитела, такое как, например, Fab, F(ab')2, одноцепочечное антитело Fab', одноцепочечное антитело Fv, моноспецифичное антитело, биспецифичное антитело, триспецифичное антитело, поливалентное антитело, химерное антитело, гуманизированное антитело, антитело человека, антитело акулы, нанотело (например, антитело, содержащее единственный мономерный вариабельный домен), антитело верблюдовых (например, из семейства Camelidae), микроантитело, интратело (например, внутриклеточное антитело) или дефукозилированное антитело и/или его производное. Миметики связывающих агентов и/или антител также находятся в рамках настоящего изобретения. Связывающий агент также может содержать присоединенную к нему детектируемую метку и/или эффекторный фрагмент.
Если связывающий агент представляет собой антигенсвязывающий белок, такой как иммуноглобулин или его производное, он может быть идентифицирован на основе нуклеотидной и/или аминокислотной последовательности, соответствующей его вариабельным областям и/или областям, определяющим комплементарность («CDR»). Например, типичный связывающий агент, который получен из описанных в настоящем документе моноклональных антител или связан с ними, может содержать тяжелую и/или легкую цепь, каждая из которых содержит одну или более константных и/или вариабельных областей. Вариабельные области обычно содержат одну или более CDR, которые в значительной степени определяют специфичность связывания антитела. Эти моноклональные антитела могут быть идентифицированы посредством анализа нуклеотидных последовательностей, кодирующих вариабельные области. Моноклональные антитела также могут быть идентифицированы посредством анализа аминокислотных последовательностей (например, кодируемых нуклеотидными последовательностями) вариабельных областей.
Аминокислоты в полифункциональных белковых молекулах согласно настоящему изобретению также могут быть заменены на любые другие аминокислоты по желанию специалиста в данной области техники. Например, специалист в данной области техники может осуществлять консервативные замены путем замены конкретных аминокислот на другие, как это известно в данной области техники. Любая из аминокислотных последовательностей антигенсвязывающих белков, описанных в настоящем документе, также может быть объединена с любой другой вариабельной областью и/или CDR в любом порядке и/или комбинации с образованием гибридных и/или слитых связывающих агентов и/или может быть вставлена в другие вариабельные области тяжелой и/или легкой цепей с использованием стандартных методик. Эти методики могут быть использованы применительно к любым константным областям.
CDR (области, определяющие комплементарность) представляют собой аминокислотные последовательности антител, которые, по меньшей мере частично, ответственны за связывание антитела со специфической мишенью. Специалистам в данной области техники понятно, что CDR могут быть идентифицированы с использованием любой из нескольких методик и/или схем. CDR связывающих агентов, приведенных в настоящем документе, могут быть идентифицированы с использованием любой из этих методик. Например, специалист в данной области техники может идентифицировать CDR с использованием схемы нумерации по Кабату, схемы нумерации по Чотиа, улучшенной схемы нумерации по Чотиа и/или любой из доступных схем определения CDR (например, AbM, контактного определения и I или описанной в источнике MacCullum, et al., J Mol. Biol., 262(5):732-745, 1996). Краткое изложение различных схем, частично основанное на, например, Kabat et al., "Sequences of Proteins of lmmunological Interest," 5th Ed., Public Health Service, National Institutes of Health, Bethesda, MD, NIH publication No. 91-3242 (1991) и Al-Lazikani et al., "Standard conformations for the canonical structures of immunoglobulins," J.Mol.Biol. 273:927-948, 1997,
Эти системы для идентификации CDR являются просто иллюстративными, и специалисту в данной области техники должно быть понятно, что могут быть подходящими другие системы. Идентифицированные таким образом CDR могут применяться для идентификации подходящих связывающих агентов, например, эквивалентов одного или более моноклональных антител, описанных в настоящем документе. Такие CDR также могут быть объединены друг с другом в любом порядке и/или комбинации с образованием гибридных и/или слитых связывающих агентов и/или вставлены в другие вариабельные области тяжелой и/или легкой цепи с использованием стандартных методик.
В некоторых вариантах реализации последовательности CDR из описанных в настоящем документе антигенсвязывающих белков присоединены к константным областям любой молекулы антитела того же самого вида (например, человека, козы, крысы, овцы, курицы), из которого была получена аминокислотная последовательность вариабельной области, или другого вида.
Дезамидирование остатков аспарагина до аспарагиновой кислоты или изоаспарагиновой кислоты является распространенной посттрансляционной модификацией белков. Дезамидирование может происходить с более высокой частотой, когда аспарагин является частью дипептида аспарагин-глицин (Asn-Gly или N-G; последовательность «NG»). Дезамидирование может оказывать неблагоприятное влияние на белки. В одном примере дезамидирование потенциально может вызывать изменение трехмерной структуры белка. В другом примере, для антитела, дезамидирование в области, которая влияет на связывание с антигеном (например, вариабельные области и/или CDR), потенциально может вызывать уменьшение или потерю связывания антитела с антигеном.
Соответственно, в некоторых вариантах реализации аминокислотные остатки, потенциально подверженные посттрансляционному дезамидированию, заменены на остатки, которые менее подвержены или не подвержены. В одном примере для модификации последовательности NG аспарагин и/или глицин заменяют, например, на любую аминокислоту, что устраняет последовательность NG.
Константные области антител получены из любого следующего источника, например, человека (например, IgG (IgG1, IgG2, IgG3, IgG4), IgM, IgA (IgA1 и IgA2), IgD и IgE), собаки (например, IgG (IgGA, IgGB, IgGC, IgGD), IgA, IgD, IgE и IgM), курицы (например, IgA, IgD, IgE, IgG, IgM, IgY), козы (например, IgG), мыши (например, IgA, IgG, IgD, IgE, IgM), свиньи (например, IgA, IgG, IgD, IgE, IgM), крысы (например, IgA, IgG, IgD, IgE, IgM), кошки (например, IgA, IgD, IgE, IgG, IgM) и/или его фрагмента и/или производного (например, в виде химерных антител).
В одном примере связывающие агенты представляют собой антитела с измененными профилями гликозилирования. Молекулы IgG, например, обычно содержат N-связанные олигосахариды. Некоторые молекулы IgG содержат олигосахарид типа двухантенарного комплекса, связанный с тяжелой цепью антитела. В IgG человека олигосахарид, обычно связан с остатком аспарагина в положении 297 (N297) тяжелой цепи (в константной Fc-области тяжелой цепи антитела). Как правило, фукоза присоединена к ближайшему к N297 остатку GLcNAC в олигосахариде. Отсутствие фукозы может увеличивать способность антител опосредовать антителозависимую клеточную цитотоксичность (АЗКЦ). Предполагается, что удаление фукозы увеличивает способность антитела взаимодействовать с Fc-рецепторами. Антитела этого типа называют «дефукозилированными». Дефукозилированные антитела могут быть получены с использованием описанных в настоящем документе методик, которые могут быть известны в данной области техники. В некоторых вариантах реализации нуклеотидная последовательность, кодирующая антитело, может быть экспрессирована в линии клеток, которые обладают измененной способностью к гликозилированию (например, удаленной, модифицированной или характеризующейся меньшим количеством фукозилтрансферазы) и не могут добавлять типичные фрагменты фукозы. Известно множество таких клеточных линий. В некоторых вариантах реализации раскрытые в настоящем документе антитела связываются с VEGF, но содержат дефукозилированные олигосахариды. Связывающие агенты (например, антитела) могут содержать другие модификации, которые могут приводить к ослаблению взаимодействия с Fc-рецепторами. Например, в молекулах антител, описанных в настоящем документе, могут быть сделаны альтернативные или дополнительные замены аминокислот.
Как описано выше, в некоторых вариантах реализации связывающие агенты могут представлять собой антитела или иммуноглобулины. Термин «антитело» или «антитела» может относиться к целым или фрагментированным антителам в неочищенной или частично очищенной форме (например, супернатант гибридомы, асцит, поликлональные антисыворотки) или в очищенной форме. «Очищенное» антитело может представлять собой антитело, которое отделено от по меньшей мере примерно 50 % белков, с которыми оно изначально обнаружено (например, в виде части супернатанта гибридомы или препарата асцита). Очищенное антитело может представлять собой антитело, которое отделено от по меньшей мере примерно 60 %, 75 %, 90 % или 95 % белков, с которыми оно изначально обнаружено. Подходящие производные также могут представлять собой фрагменты (например, Fab, F(ab')2 или одноцепочечные антитела, такие как, например, Fv). Антитела могут иметь любые подходящие происхождение или форму, включая, например, антитела мыши (например, вырабатываемые гибридомными клетками мыши), или могут быть экспрессированы в виде химерных антител, и тому подобное.
Способы получения и применения различных типов антител хорошо известны специалистам в данной области техники и могут подходить для практического применения настоящего изобретения (см., например, Harlow, et al. Antibodies: A Laboratory Manual, Cold Spring Harbor Laboratory, 1988; Harlow, et al., Using Antibodies: A Laboratory Manual, Portable Protocol No. 1, 1998; Kohler and Milstein, Nature, 256:495, 1975; Jones et al., Nature, 321:522-525, 1986; Riechmann et al., Nature, 332:323-329, 1988; Presta, Curr. Op. Struct. Biol., 2:593-596, 1992; Verhoeyen et al., Science, 239:1534-1536, 1988; Hoogenboom et al., J Mol. Biol., 227:381, 1991; Marks et al., J Mol. Biol., 222:581, 1991; Cole et al., Monoclonal Antibodies and Cancer Therapy, Alan R. Liss, p. 77, 1985; Boerner et al., J Immunol., 147(1):86-95, 1991; Marks et al., BioiTechnology 10, 779-783, 1992; Lonberg et al., Nature 368:856-859, 1994; Morrison, Nature 368:812-13, 1994; Fishwild et al., Nature Biotechnology 14, 845-51, 1996; Neuberger, Nature Biotechnology 14, 826, 1996; Lonberg and Huszar, Intern. Rev. Immunol. 13:65-93, 1995; а также патенты США №№ 4816567, 5545807, 5545806, 5569825, 5625126, 5633425 и 5661016).
В некоторых предпочтительных вариантах реализации антигенсвязывающий белок связывается с ингибирующим иммунный ответ белком контрольной точки, выбранным из группы, состоящей из PD-1, PD-L1, PD-L2, CTLA-4, CD27, CD28, CD40, CD47, CD115, CD122, CD137, OX40, GITR, ICOS, A2AR, B7-H3, B7-H4, BTLA, IDO, KIR, LAG3, NOX2, TIM-3, VISTA, SIGLEC-7, TIGIT и 4-1BB. В некоторых вариантах реализации антигенсвязывающий белок ингибирует активность ингибирующего иммунный ответ белка контрольной точки, выбранного из группы, состоящей из PD-1, PD-L1, PD-L2, CTLA-4, CD27, CD28, CD40, CD47, CD115, CD122, CD137, OX40, GITR, ICOS, A2AR, B7-H3, B7-H4, BTLA, IDO, KIR, LAG3, NOX2, TIM-3, VISTA, SIGLEC-7, TIGIT и 4-1BB. В некоторых вариантах реализации антитело представляет собой коммерчески доступное моноклональное антитело человека или гуманизированное моноклональное антитело, которое нацелено на VEGF (например, бевацизумаб (Авастин, Avastin), ранибизумаб (Луцентис, Lucentis) или пегаптаниб (Макуген, Macugen)) или молекулу контрольной точки иммунной системы, такую как CTLA-4 (например, ипилимумаб (Ервой)), PD-L1 (например, атезолизумаб (Тецентрик, Tecentriq), авелумаб (Бавенсио, Bavencio) или дурвалумаб (Имфинзи, Imfinzi)) или PD-1 (например, ниволумаб (Опдиво) или пембролизумаб (Кейтруда)).
Бевацизумаб (Авастин) представляет собой рекомбинантное гуманизированное моноклональное антитело, которое блокирует ангиогенез за счет ингибирования фактора роста эндотелия сосудов A (VEGF-A). VEGF-A представляет собой белковый фактор роста, который стимулирует ангиогенез при различных заболеваниях, в частности, при раке. Бевацизумаб являлся первым доступным ингибитором ангиогенеза в США.
Ипилимумаб (Ервой) представляет собой моноклональное антитело, которое активирует иммунную систему за счет нацеливания на CTLA-4, подавляющий функцию иммунной системы белковый рецептор, и классифицируется как лекарственное средство-ингибитор контрольной точки. T-лимфоциты могут распознавать и разрушать раковые клетки. Однако ингибиторный механизм препятствует этому разрушению. Ипилимумаб блокирует этот ингибиторный механизм и обеспечивает дальнейшее разрушение раковых клеток лимфоцитами. Раковые клетки вырабатывают антигены, которые могут быть использованы иммунной системой для их идентификации. Эти антигены распознаются дендритными клетками, которые презентируют антигены цитотоксическим T-лимфоцитам (CTL) в лимфатических узлах. CTL распознают раковые клетки по этим антигенам и разрушают их. Однако наряду с антигенами дендритные клетки презентируют ингибиторную сигнальную молекулу. Эта сигнальная молекула связывается с рецептором, ассоциированным с цитотоксическими T-лимфоцитами антигеном 4 (CTLA-4), на CTL и прекращает цитотоксическую реакцию. Это обусловливает выживание раковых клеток. Ипилимумаб связывается с CTLA-4, блокируя ингибирующий сигнал, что обеспечивает разрушение раковых клеток под действием CTL.
Атезолизумаб (Тецентрик) представляет собой полностью гуманизированное конструированное моноклональное антитело изотипа IgG1 против белка PD-L1. Авелумаб (Бавенсио) представляет собой полностью человеческое моноклональное антитело, которое связывается с PD-L1. Дурвалумаб (Имфинзи) представляет собой полностью человеческое моноклональное антитело, которое связывается с PD-L1. Вместе эти молекулы могут быть названы ингибиторами PD-L1 и классифицированы как ингибиторы иммунных контрольных точек. PD-L1 может экспрессироваться на высоком уровне на некоторых опухолях, что приводит к уменьшению активации иммунных клеток (в частности, цитотоксических T-клеток), которые в противном случае могли бы распознавать и атаковать раковые клетки. Связывающие PD-L1 ингибиторы контрольных точек блокируют взаимодействие PD-L1 с белком 1 программируемой гибели клеток (PD-1) и рецепторами CD80 (B7-1R). Ингибирование PD-L1 устраняет подавление функции иммунной системы и таким образом вызывает противоопухолевый ответ.
Ниволумаб (Опдиво) представляет собой моноклональное антитело человека, которое связывается с PD-1. Пембролизумаб (Кейтруда) представляет собой гуманизированное антитело, которое связывается с PD-1. Вместе эти молекулы могут быть названы ингибиторами PD-1 и классифицированы как ингибиторы иммунных контрольных точек. Эти молекулы действуют путем блокирования отрицательного регулятора активации T-клеток, которое позволяет иммунной системе атаковать опухоль. Это является примером блокирования иммунной контрольной точки. PD-1 представляет собой белок на поверхности активированных T-клеток. Как обсуждалось выше, если PD-L1 или PD-L2 связываются с PD-1, T-клетка становится неактивной. Многие раковые клетки вырабатывают PD-L1, который препятствует атаке T-клеток на опухоли. Ниволумаб блокирует связывание PD-L1 с PD-1, обеспечивая функционирование T-клеток.
Специалистам в данной области техники будет понятно, что CDR или вариабельные области упомянутых антител могут быть выделены (например, путем клонирования) и введены в другие каркасные области или производные антигенсвязывающих белков (например, Fab, F(ab')2, одноцепочечное антитело Fab', одноцепочечное антитело Fv, биспецифичное антитело, триспецифичное антитело, поливалентное антитело, гуманизированное антитело, нанотело, антитело верблюдовых, микроантитело или интратело) по желанию. Настоящее изобретение охватывает антигенсвязывающие белки, которые получены из эталонных антител и которые идентифицированы на основе CDR вариабельных областей эталонных антител. Например, в некоторых предпочтительных вариантах реализации антигенсвязывающие белки согласно настоящему изобретению содержат вариабельные области тяжелой и легкой цепей из бевацизумаба, ранибизумаба, пегаптаниба, ипилимумаба, атезолизумаба, авелумаба, дурвалумаба, ниволумаба или пембролизумаба. В других предпочтительных вариантах реализации антигенсвязывающие белки содержат один, два или все три из CDR1, CDR2 и CDR3 из вариабельных областей тяжелой и легкой цепей из бевацизумаба, ранибизумаба, пегаптаниба, ипилимумаба, атезолизумаба, авелумаба, дурвалумаба, ниволумаба или пембролизумаба.
III. Полифункциональные полипептиды
В вариантах реализации настоящего изобретения предложены полифункциональные полипептиды и/или полинуклеотиды, кодирующие гибридный полипептид, содержащий полипептид декорина, функционально связанный со связывающим агентом. В некоторых вариантах реализации также предложены композиции, содержащие эти связывающие агенты, полипептиды, пептиды, полинуклеотиды, векторы экспрессии и/или клетки-хозяева. В некоторых вариантах реализации композиции содержат фармацевтически приемлемый носитель.
В некоторых предпочтительных вариантах реализации связывающий агент в полифункциональной белковой молекуле представляет собой антитело, описанное выше. Схематическое изображение гибридного белка декорин-антитело согласно настоящему изобретению, нацеленного на VEGF, представлено на ФИГ. 1. Квалифицированному специалисту в данной области техники будет понятно, что другие антигенсвязывающие белки, описанные в настоящем документе, могут заменить антитело к VEGF, описанное на ФИГ. 1. Как показано на ФИГ. 1, гибридный белок содержит тяжелую и легкую цепи антитела, которое связывается с VEGF (например, бевацизумаба). Молекула декорина функционально связана с C-концом каждой тяжелой цепи посредством пептидного линкера. Настоящее изобретение не ограничивается применением какого-либо конкретного пептидного линкера или связыванием молекулы декорина с какой-либо конкретной аминокислотой антигенсвязывающего белка. В некоторых предпочтительных вариантах реализации молекула декорина связана через C-конец одной или двух тяжелых цепей антитела, через C-конец одной или двух легких цепей антитела, через N-конец одной или двух тяжелых цепей антитела, через N-конец одной или двух легких цепей антитела, либо через аминокислоту в константной области одной или двух тяжелых цепей антитела, которая химически модифицирована для обеспечения присоединения полипептидов, таких как молекула декорина. Соответственно, гибридные белки согласно настоящему изобретению могут содержать одну или предпочтительно две молекулы декорина или их функциональные участки и могут содержать более двух молекул декорина или их функциональных участков.
В примерах представлен ряд конструкций нуклеиновых кислот, кодирующих полифункциональные белковые молекулы согласно настоящему изобретению. Последовательности кратко приведены в следующей таблице.
Таким образом, в некоторых предпочтительных вариантах реализации гибридные белки согласно настоящему изобретению содержат вариабельные области тяжелой и легкой цепей из бевацизумаба, ранибизумаба, пегаптаниба, ипилимумаба, атезолизумаба, авелумаба, дурвалумаба, ниволумаба или пембролизумаба, функционально связанные с молекулой декорина, предпочтительно с коровым белком декорина. В других предпочтительных вариантах реализации гибридные белки содержат один, два или все три из CDR1, CDR2 и CDR3 из вариабельных областей тяжелой и легкой цепей из бевацизумаба, ранибизумаба, пегаптаниба, ипилимумаба, атезолизумаба, авелумаба, дурвалумаба, ниволумаба или пембролизумаба, функционально связанных с молекулой декорина, предпочтительно коровым белком декорина.
В некоторых предпочтительных вариантах реализации молекула декорина (или молекулы, если применяют более одной копии) представляет собой коровый белок декорина, который на по меньшей мере 80 %, 90 %, 95 %, 99 % или 100 % идентичен SEQ ID NO:7. В других предпочтительных вариантах реализации молекула декорина (или молекулы, если применяют более одной копии) представляет собой связывающий TGF-β домен декорина, который на по меньшей мере 80 %, 90 %, 95 %, 99 % или 100 % идентичен SEQ ID NO:64 или 66. В некоторых предпочтительных вариантах реализации коровый белок декорина ингибирует активность TGF-β. В некоторых предпочтительных вариантах реализации коровый белок декорина содержит одну или более мутаций, которые вызывают отсутствие гагилирования корового белка декорина. В некоторых предпочтительных вариантах реализации коровый белок декорина содержит мутацию в положении аминокислоты 4 (то есть 4ой аминокислоты от N-конца) молекулы корового белка зрелого декорина. В некоторых предпочтительных вариантах реализации молекула корового белка декорина связана с тяжелой или легкой цепью целевого антитела посредством линкерной последовательности. Настоящее изобретение не ограничивается какой-либо конкретной линкерной последовательностью. В некоторых предпочтительных вариантах реализации линкерная последовательность представляет собой SEQ ID NO:6. В некоторых особенно предпочтительных вариантах реализации линкерная последовательность присоединена к N-концу тяжелой цепи антитела и расположена между тяжелой цепью и коровым белком декорина. Соответственно, в некоторых предпочтительных вариантах реализации гибридная молекула, содержащая тяжелую цепь, может быть представлена следующей формулой:
Белок тяжелой цепи - линкер - коровый белок декорина
В других предпочтительных вариантах реализации гибридные молекулы могут быть представлены следующими формулами:
C-конец белка тяжелой цепи - линкер - декорин
C-конец белка легкой цепи - линкер - декорин
декорин - линкер - N-конец белка тяжелой цепи
декорин - линкер - N-конец белка легкой цепи
Константная область - линкер - декорин
где декорин может представлять собой декорин дикого типа, коровый белок декорина или его функциональный участок, как подробно описано выше, и где соединение осуществляется посредством амидных связей, характерных для гибридного белка, как известно в данной области техники, или с помощью химически модифицированной аминокислоты в N-концевой, C-концевой или константной области антигенсвязывающего белка. Например, меченные альдегидом полипептиды иммуноглобулина (Ig) могут быть преобразованы под действием фермента, участвующего в образовании формилглицина, с получением модифицированного полипептида Ig, содержащего 2-формилглицин (FGly). Затем FGly-модифицированный полипептид Ig может быть ковалентно и сайт-специфично связан с целевым фрагментом с получением конъюгата Ig. См., например, патент США № 10183998, который полностью включен в настоящий документ посредством ссылки.
В некоторых предпочтительных вариантах реализации секреторная сигнальная последовательность предшествует последовательности белка тяжелой цепи для обеспечения секреции из клеток-хозяев в процессе выработки белка. Аналогичным образом, в некоторых предпочтительных вариантах реализации секреторная сигнальная последовательность предшествует последовательности белка легкой цепи для обеспечения секреции из клеток-хозяев в процессе выработки белка.
В некоторых предпочтительных вариантах реализации, где нацеливающая молекула представляет собой бевацизумаб, последовательность тяжелой цепи на по меньшей мере 90 %, 95 %, 99 % или 100 % идентична SEQ ID NO:8, а последовательность легкой цепи на по меньшей мере 90 %, 95 %, 99 % или 100 % идентична SEQ ID NO:9. В некоторых предпочтительных вариантах реализации нацеливающая молекула связывается с VEGF-A или ингибирует его.
В некоторых предпочтительных вариантах реализации, где нацеливающая молекула представляет собой ипилимумаб, последовательность тяжелой цепи на по меньшей мере 90 %, 95 %, 99 % или 100 % идентична SEQ ID NO:14, а последовательность легкой цепи на по меньшей мере 90 %, 95 %, 99 % или 100 % идентична SEQ ID NO:15. В некоторых предпочтительных вариантах реализации нацеливающая молекула связывается с CTLA-4 или ингибирует его.
В некоторых предпочтительных вариантах реализации, где нацеливающая молекула представляет собой атезолизумаб, последовательность тяжелой цепи на по меньшей мере 90 %, 95 %, 99 % или 100 % идентична SEQ ID NO:20, а последовательность легкой цепи на по меньшей мере 90 %, 95 %, 99 % или 100 % идентична SEQ ID NO:21. В некоторых предпочтительных вариантах реализации нацеливающая молекула связывается с PD-L1 или ингибирует его.
В некоторых предпочтительных вариантах реализации, где нацеливающая молекула представляет собой авелумаб, последовательность тяжелой цепи на по меньшей мере 90 %, 95 %, 99 % или 100 % идентична SEQ ID NO:26, а последовательность легкой цепи на по меньшей мере 90 %, 95 %, 99 % или 100 % идентична SEQ ID NO:27. В некоторых предпочтительных вариантах реализации нацеливающая молекула связывается с PD-L1 или ингибирует его.
В некоторых предпочтительных вариантах реализации, где нацеливающая молекула представляет собой дурвалумаб, последовательность тяжелой цепи на по меньшей мере 90 %, 95 %, 99 % или 100 % идентична SEQ ID NO:32, а последовательность легкой цепи на по меньшей мере 90 %, 95 %, 99 % или 100 % идентична SEQ ID NO:33. В некоторых предпочтительных вариантах реализации нацеливающая молекула связывается с PD-L1 или ингибирует его.
В некоторых предпочтительных вариантах реализации, где нацеливающая молекула представляет собой ниволумаб, последовательность тяжелой цепи на по меньшей мере 90 %, 95 %, 99 % или 100 % идентична SEQ ID NO:38, а последовательность легкой цепи на по меньшей мере 90 %, 95 %, 99 % или 100 % идентична SEQ ID NO:39. В некоторых предпочтительных вариантах реализации нацеливающая молекула связывается с PD-1 или ингибирует его.
В некоторых предпочтительных вариантах реализации, где нацеливающая молекула представляет собой пембролизумаб, последовательность тяжелой цепи на по меньшей мере 90 %, 95 %, 99 % или 100 % идентична SEQ ID NO:44, а последовательность легкой цепи на по меньшей мере 90 %, 95 %, 99 % или 100 % идентична SEQ ID NO:45. В некоторых предпочтительных вариантах реализации нацеливающая молекула связывается с PD-1 или ингибирует его.
В некоторых вариантах реализации предложены препараты гибридных белков. Такие препараты могут содержать, например, неочищенные или очищенные гибридные белки. Как правило, такие препараты содержат буфер, такой как фосфатно-солевой буфер или трис-солевой буфер (ФСБ или ТСБ, соответственно). Приготовленные препараты также могут содержать вспомогательные вещества, например, такие как стабилизаторы.
В некоторых предпочтительных вариантах реализации настоящего изобретения дополнительно предложены конструкции для экспрессии нуклеиновых кислот, которые кодируют гибридные белки согласно настоящему изобретению. Соответственно, в некоторых вариантах реализации экспрессионные конструкции кодируют последовательности гибридных белков, описанные выше и функционально связанные с дополнительными нуклеотидными последовательностями, необходимыми для экспрессии в выбранной системе экспрессии.
В одном примере нуклеотидные последовательности, кодирующие гибридный белок, встраивают в векторную систему и затем экспрессируют в клетках-хозяевах. В одном примере клетки-хозяева представляют собой культивируемые клетки. В одном примере векторную систему применяют в культивируемых клетках млекопитающих в условиях, при которых происходит экспрессия гибридных белков.
Гибридные полинуклеотиды согласно настоящему изобретению могут применяться для получения гибридных полипептидов рекомбинантными способами. Таким образом, например, полинуклеотид может быть включен в любой из множества векторов экспрессии для экспрессии полипептида. В некоторых вариантах реализации настоящего изобретения векторы включают, но не ограничиваются указанными, ретровирусные векторы, хромосомные, нехромосомные и синтетические последовательности ДНК (например, производные SV40, бактериальные плазмиды, фаговую ДНК; бакуловирус, дрожжевые плазмиды, векторы, полученные из комбинаций плазмиды и фаговой ДНК, и ДНК вирусов, таких как вирус коровьей оспы, аденовирус, вирус оспы птиц и вирус псевдобешенства). Предполагается, что может быть использован любой вектор, если он является реплицируемым и жизнеспособным в организме-хозяине. В некоторых предпочтительных вариантах реализации векторы представляют собой ретровирусные векторы, описанные в патентах США №№ 6852510 и 7332333 и публикациях на патенты США №№ 200402335173 и 20030224415, все из которых полностью включены в настоящий документ посредством ссылок. В некоторых особенно предпочтительных вариантах реализации векторы представляют собой псевдотипированные ретровирусные векторы.
В частности, в некоторых вариантах реализации настоящего изобретения предложены рекомбинантные конструкции, содержащие одну или более последовательностей, как описано выше в общих чертах. В некоторых вариантах реализации настоящего изобретения указанные конструкции содержат вектор, такой как плазмидный или вирусный вектор, в который была вставлена последовательность согласно настоящему изобретению, в прямой или обратной ориентации. В других вариантах реализации гетерологичная структурная последовательность собрана в подходящей фазе с последовательностями инициации и терминации трансляции. В предпочтительных вариантах реализации настоящего изобретения подходящую последовательность ДНК вставляют в вектор с использованием любой из множества методик. Как правило, последовательность ДНК вставляют в подходящий(ие) сайт(ы) для эндонуклеаз рестрикции с помощью методик, известных в данной области техники.
Большое количество подходящих векторов известны специалистам в данной области техники и являются коммерчески доступными. Такие векторы включают, но не ограничиваются указанными, следующие векторы: 1) бактериальные - pQE70, pQE60, pQE-9 (Qiagen), pBS, pD10, Phagescript, psiX174, Bluescript SK, pBSKS, pNH8A, pNH16a, pNH18A, pNH46A (Stratagene); ptrc99a, pKK223-3, pKK233-3, pDR540, pRIT5 (Pharmacia); 2) эукариотические - pWLNEO, pSV2CAT, pOG44, PXT1, pSG (Stratagene), pSVK3, pBPV, pMSG, pSVL (Pharmacia); и 3) бакуловирусные - pPbac и pMbac (Stratagene). Могут быть использованы любые другие плазмида или вектор, если они являются реплицируемыми и жизнеспособными в организме-хозяине. В некоторых предпочтительных вариантах реализации настоящего изобретения векторы экспрессии млекопитающих содержат точку начала репликации, подходящий промотор и энхансер, а также любые необходимые сайты связывания рибосом, сайты полиаденилирования, донорные и акцепторные сайты сплайсинга, последовательности терминации транскрипции и 5'-фланкирующие нетранскрибируемые последовательности. В других вариантах реализации последовательности ДНК, полученные из сайтов сплайсинга и полиаденилирования SV40, могут быть использованы для обеспечения необходимых нетранскрибируемых генетических элементов.
В некоторых вариантах реализации настоящего изобретения последовательность ДНК в векторе экспрессии функционально связана с соответствующей последовательностью(ями), регулирующей экспрессию (промотором), для управления синтезом мРНК. Подходящие для целей настоящего изобретения промоторы включают, но не ограничиваются указанными, промотор LTR или SV40, lac или trp E. coli, PL и PR фага лямбда, промоторы T3 и T7 и немедленно-ранний промотор цитомегаловируса (CMV), промоторы тимидинкиназы простого вируса герпеса (HSV) и металлотионеина-I мыши и другие промоторы, которые, как известно, регулируют экспрессию генов в прокариотических или эукариотических клетках или их вирусах. В других вариантах реализации настоящего изобретения рекомбинантные векторы экспрессии содержат точки начала репликации и селектируемые маркеры, обеспечивающие трансформацию клетки-хозяина (например, устойчивость к дигидрофолатредуктазе или неомицину для культуры эукариотических клеток, или устойчивость к тетрациклину или ампициллину для E. coli).
В некоторых вариантах реализации настоящего изобретения транскрипцию ДНК, кодирующей полипептиды согласно настоящему изобретению, в высших эукариотах увеличивают путем вставки энхансерной последовательности в вектор. Энхансеры представляют собой цис-действующие элементы ДНК, обычно имеющие размер от 10 до 300 п.о., которые действуют на промотор, увеличивая его транскрипцию. Подходящие для целей настоящего изобретения энхансеры включают, но не ограничиваются указанными, энхансер SV40 на расстоянии от 100 до 270 п. о. после точки начала репликации, энхансер немедленно-раннего промотора цитомегаловируса, энхансер полиомы после точки начала репликации и энхансеры аденовируса.
В других вариантах реализации вектор экспрессии также содержит сайт связывания рибосомы для инициации трансляции и терминатор транскрипции. В других вариантах реализации настоящего изобретения вектор также может содержать подходящие последовательности для амплификации экспрессии.
В другом варианте реализации настоящего изобретения предложены клетки-хозяева, содержащие описанные выше конструкции. В некоторых вариантах реализации настоящего изобретения клетка-хозяин представляет собой клетку высших эукариот (например, клетку млекопитающего или насекомого). В других вариантах реализации настоящего изобретения клетка-хозяин представляет собой клетку низших эукариот (например, клетку дрожжей). В других вариантах реализации настоящего изобретения клетка-хозяин может представлять собой прокариотическую клетку (например, бактериальную клетку). Конкретные примеры клеток-хозяев включают, но не ограничиваются указанными, Escherichia coli, Salmonella typhimurium, Bacillus subtilis и различные виды, относящиеся к родам Pseudomonas, Streptomyces и Staphylococcus, а также Saccharomyces cerivisiae, Schizosaccharomyces pombe, клетки Drosophila S2, клетки Spodoptera Sf9, клетки яичника китайского хомячка (CHO), линии COS-7 фибробластов почки обезьяны (Gluzman, Cell 23:175 [1981]), линии клеток C127, 3T3, 293, 293T, HeLa и BHK.
Конструкции в клетках-хозяевах могут применяться обычным образом для получения генного продукта, кодируемого рекомбинантной последовательностью. В некоторых вариантах реализации введение конструкции в клетку-хозяина может быть выполнено посредством ретровирусной трансдукции, трансфекции с применением фосфата кальция, опосредованной ДЭАЭ-декстраном трансфекции или электропорации (см., например, Davis et al. [1986] Basic Methods in Molecular Biology). В качестве альтернативы, в некоторых вариантах реализации настоящего изобретения полипептиды согласно настоящему изобретению могут быть получены путем синтеза с помощью обычных синтезаторов пептидов.
Белки могут быть экспрессированы в клетках млекопитающих, дрожжах, бактериях или других клетках под контролем подходяших промоторов. Бесклеточные системы трансляции также могут применяться для получения таких белков с использованием РНК, полученных из конструкций ДНК согласно настоящему изобретению. Подходящие для использования с прокариотическими и эукариотическими хозяевами векторы клонирования и экспрессии описаны в источнике Sambrook, et al. (1989) Molecular Cloning: A Laboratory Manual, Second Edition, Cold Spring Harbor, N.Y.
В некоторых вариантах реализации настоящего изобретения после трансформации подходящего штамма-хозяина и выращивания штамма-хозяина в среде до соответствующей плотности клеток происходит секреция белка, и клетки культивируют в течение дополнительного периода времени. В других вариантах реализации настоящего изобретения клетки, как правило, собирают посредством центрифугирования, разрушают с применением физических или химических методов, и полученный неочищенный экстракт оставляют для дальнейшей очистки. В других вариантах реализации настоящего изобретения клетки микроорганизмов, используемые для экспрессии белков, могут быть разрушены с применением любого удобного способа, включая циклы замораживания-размораживания, обработку ультразвуком, механическое разрушение или использование лизирующих клетки агентов.
В дополнительных вариантах реализации предложены наборы, содержащие гибридные белки и, необязательно, другие компоненты, которые являются подходящими, необходимыми или достаточными для применения гибридных белков (например, применения в терапевтических, исследовательских и скрининговых целях). Гибридные белки набора могут быть обеспечены в любой подходящей форме, включая замороженную, лиофилизированную, либо в фармацевтически приемлемом буфере, таком как ТСБ или ФСБ.
Описанные в настоящем документе гибридные белки и/или их производные также могут быть включены в композиции для применения in vitro или in vivo. Антитело, гибридный белок или их производные также могут быть прочно присоединены к функциональным эффекторным фрагментам, таким как цитотоксические лекарственные средства или токсины или их активные фрагменты, такие как, среди прочего, A-цепь дифтерийного токсина, A-цепь экзотоксина, A-цепь рицина, A-цепь абрина, курцин, кротин, феномицин, эномицин.
Функциональные фрагменты также могут содержать радионуклиды. В одном варианте реализации эффекторные фрагменты могут быть прочно присоединены к связывающим агентам. В одном примере детектируемые метки прочно присоединены к связывающим агентам посредством химических связей. В одном примере химические связи представляют собой ковалентные химические связи. В одном примере эффекторные фрагменты конъюгированы со связывающими агентами.
Описанные в настоящем документе гибридные белки могут быть приготовлены в виде препарата для инъекций, например, в виде суспензии в нетоксичном парентерально приемлемом разбавителе или растворителе. Подходящие переносящие среды и растворители, которые могут применяться, включают, среди прочего, воду, раствор Рингера и изотонический раствор хлорида натрия, ТСБ и ФСБ. Составы могут содержать вспомогательные вещества, такие как, например, стабилизаторы. В некоторых приложениях антитела подходят для применения in vitro. В других приложениях антитела подходят для применения in vivo. Препараты, подходящие для применения в обоих случаях, хорошо известны в данной области техники и будут меняться в зависимости от конкретного применения.
Гибридные белки могут быть объединены с одним или более фармацевтически приемлемыми носителями перед введением в организм-хозяин. Фармацевтически приемлемый носитель представляет собой материал, который не является нежелательным с биологической или иной точки зрения, например, этот материал можно вводить субъекту, не вызывая при этом каких-либо нежелательных биологических эффектов или неблагоприятного взаимодействия с любым из других компонентов содержащей его фармацевтической композиции. Носитель, несомненно, должен быть выбран таким образом, чтобы минимизировать любую деградацию активного ингредиента и чтобы минимизировать любые неблагоприятные побочные эффекты у субъекта, что должно быть хорошо известно специалисту в данной области техники. Подходящие фармацевтические носители и их составы описаны, например, в источнике Remington’s: The Science and Practice of Pharmacy, 2 yt Edition, David B. Troy, ed., Lippicott Williams & Wilkins (2005). Как правило, в составе используют подходящее количество фармацевтически приемлемой соли для того, чтобы композиция стала изотонической. Примеры фармацевтически приемлемых носителей включают, но не ограничиваются указанными, стерильную воду, солевой раствор, буферные растворы, такие как раствор Рингера, и декстрозу. pH раствора обычно составляет от примерно 5 до примерно 8 или от примерно 7 до примерно 7,5. Другие носители включают препараты с замедленным высвобождением, такие как полупроницаемые матрицы из твердых гидрофобных полимеров, содержащие полипептиды или их фрагменты. Матрицы могут находиться в форме изделий определенной формы, например, пленок, липосом или микрочастиц. Специалистам в данной области техники будет очевидно, что некоторые носители могут быть более предпочтительными в зависимости от, например, пути введения и концентрации вводимой композиции. Носители представляют собой носители, подходящие для введения полипептидов и/или их фрагментов людям или другим субъектам.
Фармацевтические композиции также могут содержать, в дополнение к гибридному белку, носители, загустители, разбавители, буферы, консерванты, поверхностно-активные агенты, адъюванты, иммуностимуляторы. Фармацевтические композиции также могут содержать один или более активных ингредиентов, таких как противомикробные агенты, противовоспалительные агенты и анестетики.
IV. Способы применения
В вариантах реализации настоящего раскрытия предложены композиции и способы исследования, скрининга и терапевтического применения. Например, в вариантах реализации настоящего изобретения предложены способы лечения различных заболеваний с применением многофункциональных белковых молекул, описанных в настоящем документе.
В некоторых вариантах реализации композиции и способы согласно настоящему изобретению применяют для лечения пораженных заболеванием клеток, тканей, органов или патологических состояний и/или заболеваний у животного (например, пациента-млекопитающего, включая, но не ограничиваясь ими, людей и животных, подлежащих ветеринарному лечению). В связи с этим различные заболевания и патологические состояния поддаются лечению или предотвращению с применением настоящих способов и композиций. Неограничивающий перечень примеров этих заболеваний и состояний включает, но не ограничивается ими, рак поджелудочной железы, рак молочной железы, рак предстательной железы, лимфому, рак кожи, рак толстой кишки, меланому, злокачественную меланому, рак яичников, рак головного мозга, первичную карциному головного мозга, рак головы и шеи, глиому, глиобластому, рак печени, рак мочевого пузыря, немелкоклеточный рак легкого, карциному головы или шеи, карциному молочной железы, карциному яичников, карциному легкого, мелкоклеточную карциному легкого, опухоль Вильмса, карциному шейки матки, карциному яичек, карциному мочевого пузыря, карциному поджелудочной железы, карциному желудка, карциному толстой кишки, карциному предстательной железы, карциному органов мочеполовой системы, карциному щитовидной железы, карциному пищевода, миелому, множественную миелому, карциному надпочечников, почечно-клеточную карциному, карциному эндометрия, карциному коры надпочечников, злокачественную инсулиному поджелудочной железы, злокачественную карциноидную опухоль, хориокарциному, грибовидный микоз, злокачественную гиперкальциемию, цервикальную гиперплазию, лейкоз, острый лимфоцитарный лейкоз, хронический лимфоцитарный лейкоз, острый миелогенный лейкоз, хронический миелогенный лейкоз, хронический гранулоцитарный лейкоз, острый гранулоцитарный лейкоз, волосатоклеточный лейкоз, нейробластому, рабдомиосаркому, саркому Капоши, истинную полицитемию, эссенциальный тромбоцитоз, болезнь Ходжкина, неходжкинскую лимфому, саркому мягких тканей, остеогенную саркому, первичную макроглобулинемию и ретинобластому и тому подобное, а также макулярную дегенерацию.
В некоторых вариантах реализации настоящего изобретения предложены способы введения эффективного количества гибридного полипептида согласно настоящему изобретению и по меньшей мере одного дополнительного терапевтического агента (включая, но не ограничиваясь ими, химиотерапевтические противоопухолевые агенты, модулирующие апоптоз агенты, противомикробные, противовирусные, противогрибковые и противовоспалительные агенты) и/или применения терапевтической методики (например, хирургического вмешательства и/или лучевой терапии). В конкретном варианте реализации дополнительный терапевтический агент (агенты) представляет собой противораковый агент.
Предполагается, что в способах согласно настоящему изобретению будет применен ряд подходящих противораковых агентов. Действительно, настоящее изобретение предполагает, но не ограничивается этим, введение множества противораковых агентов, таких как: агенты, индуцирующие апоптоз; полинуклеотиды (например, антисмысловые, рибозимы, миРНК); полипептиды (например, ферменты и антитела); биологические миметики; алкалоиды; алкилирующие агенты; противоопухолевые антибиотики; антиметаболиты; гормоны; соединения платины; моноклональные или поликлональные антитела (например, антитела, конъюгированные с противораковыми лекарственными средствами, токсинами, дефенсинами), токсины; радионуклиды; модификаторы биологического ответа (например, интерфероны (например, ИФН-α) и интерлейкины (например, IL-2)); агенты для адоптивной иммунотерапии; гематопоэтические факторы роста; агенты, индуцирующие дифференцировку опухолевых клеток (например, полностью транс-ретиноевая кислота); агенты для генной терапии (например, агенты для антисмысловой терапии и нуклеотиды); противоопухолевые вакцины; ингибиторы ангиогенеза; ингибиторы протеасомы: модуляторы NF-КB; анти-CDK соединения; ингибиторы HDAC и тому подобное. Множество других примеров химиотерапевтических соединений и противораковых терапевтических агентов, подходящих для совместного введения с раскрытыми соединениями, известны специалистам в данной области техники.
В некоторых вариантах реализации противораковые агенты включают агенты, индуцирующие или стимулирующие апоптоз. Агенты, индуцирующие апоптоз, включают, но не ограничиваются указанными, излучение (например, рентгеновское излучение, гамма-излучение, УФ); родственные фактору некроза опухоли (TNF) факторы (например, белки семейства рецепторов TNF, лиганды семейства TNF, TRAIL, антитела к TRAIL-R1 или TRAIL-R2); ингибиторы киназ (например, ингибитор киназы рецептора эпидермального фактора роста (EGFR), ингибитор киназы рецептора сосудистого фактора роста (VGFR), ингибитор киназы рецептора фактора роста фибробластов (FGFR), ингибитор киназы рецептора тромбоцитарного фактора роста (PDGFR) и ингибиторы киназы Bcr-Abl (такие как Гливек (GLEEVEC)); антисмысловые молекулы; антитела (например, Герцептин (HERCEPTIN), Ритуксан (RITUXAN), Зевалин (ZEVALIN) и Авастин (AVASTIN)); антиэстрогены (например, ралоксифен и тамоксифен); антиандрогены (например, флутамид, бикалутамид, финастерид, аминоглутетимид, кетоконазол и кортикостероиды); ингибиторы циклооксигеназы 2 (ЦОГ-2) (например, целекоксиб, мелоксикам, NS-398 и нестероидные противовоспалительные средства (НПВС)); противовоспалительные лекарственные средства (например, бутазолидин, Декадрон (DECADRON), Дельтазон (DELTASONE), дексаметазон, Дексаметазон Интенсол (Dexamethasone Intensol), Дексон (DEXONE), Гексадрол (HEXADROL), гидроксихлорохин, Метикортен (METICORTEN), Орадексон (ORADEXON), Оразон (ORASONE), оксифенбутазон, Педиапред (PEDIAPRED), фенилбутазон, Плаквенил (PLAQUENIL), преднизолон, преднизон, Прелон PRELONE и Тандеарил (TANDEARIL)); а также противораковые химиотерапевтические лекарственные средства (например, иринотекан (Камптозар (CAMPTOSAR)), CPT-11, флударабин (Флудара (FLUDARA)), дакарбазин (DTIC), дексаметазон, митоксантрон, Милотарг (MYLOTARG), VP-16, цисплатин, карбоплатин, оксалиплатин, 5-ФУ (5-FU), доксорубицин, гемцитабин, бортезомиб, гефитиниб, бевацизумаб, Таксотер (TAXOTERE) или Таксол (TAXOL)); клеточные сигнальные молекулы; церамиды и цитокины; стауроспорин и тому подобное.
В других вариантах реализации в композициях и способах согласно настоящему изобретению предложены соединение согласно настоящему изобретению и по меньшей мере один антигиперпролиферативный или противоопухолевый агент, выбранный из алкилирующих агентов, антиметаболитов и природных продуктов (например, соединений, полученных из трав и других растений и/или животных).
Алкилирующие агенты, подходящие для применения в настоящих композициях и способах, включают, но не ограничиваются указанными: 1) производные азотистого иприта (например, мехлорэтамин, циклофосфамид, ифосфамид, мелфалан (L-сарколизин) и хлорамбуцил); 2) этиленимины и метилмеламины (например, гексаметилмеламин и тиотепу); 3) алкилсульфонаты (например, бусульфан); 4) производные нитрозомочевины (например, кармустин (BCNU); ломустин (CCNU); семустин (метил-CCNU) и стрептозоцин (стрептозотоцин)) и 5) триазены (например, дакарбазин (DTIC; диметилтриазеноимид-азолкарбоксамид).
В некоторых вариантах реализации антиметаболиты, подходящие для применения в настоящих композициях и способах, включают, но не ограничиваются указанными: 1) аналоги фолиевой кислоты (например, метотрексат (аметоптерин)); 2) аналоги пиримидина (например, фторурацил (5-фторурацил; 5-FU), флоксуридин (фтордезоксиуридин; FudR) и цитарабин (цитозина арабинозид)); и 3) аналоги пурина (например, меркаптопурин (6-меркаптопурин; 6-MP), тиогуанин (6-тиогуанин; TG) и пентостатин (2’-дезоксикоформицин)).
В других вариантах реализации химиотерапевтические агенты, подходящие для применения в композициях и способах согласно настоящему изобретению, включают, но не ограничиваются указанными: 1) алкалоиды барвинка (например, винбластин (VLB), винкристин); 2) эпиподофиллотоксины (например, этопозид и тенипозид); 3) антибиотики (например, дактиномицин (актиномицин D), даунорубицин (дауномицин; рубидомицин), доксорубицин, блеомицин, пликамицин (митрамицин) и митомицин (митомицин C)); 4) ферменты (например, L-аспарагиназу); 5) модификатор биологического ответа (например, интерферон альфа); 6) координационные комплексы платины (например, цисплатин (цис-DDP) и карбоплатин); 7) антрацендионы (например, митоксантрон); 8) замещенные мочевины (например, гидроксимочевину); 9) производные метилгидразина (например, прокарбазин (N-метилгидразин; MIH)); 10) ингибиторы синтеза гормонов коры надпочечников (например, митотан (o,p’-DDD) и аминоглутетимид); 11) адренокортикостероиды (например, преднизон); 12) прогестины (например, гидроксипрогестерона капроат, медроксипрогестерона ацетат и мегестрола ацетат); 13) эстрогены (например, диэтилстильбэстрол и этинилэстрадиол); 14) антиэстрогены (например, тамоксифен); 15) андрогены (например, тестостерона пропионат и флюоксиместерон); 16) антиандрогены (например, флутамид): и 17) аналоги гонадотропин-рилизинг гормона (например, лейпорид).
Любой онколитический агент, который обычно применяют в контексте терапии рака, находит применение в композициях и способах согласно настоящему изобретению. Например, Управление по контролю за пищевыми продуктами и лекарственными средствами США (U.S. Food and Drug Administration, U.S.F.D.A.) ведет формуляр онколитических агентов, одобренных для применения в Соединенных Штатах. Международные организации, аналогичные U.S.F.D.A., ведут схожие формуляры. В таблице ниже представлен перечень иллюстративных противоопухолевых агентов, одобренных для применения в Соединенных Штатах. Специалистам в данной области техники будет понятно, что в «инструкциях по применению», необходимых для всех одобренных в США химиотерапевтических средств, описаны утвержденные показания, информация о дозировании, данные о токсичности и тому подобное для указанных иллюстративных агентов.
(дез-аланил-1, серин-125 интерлейкин-2 человека)
(антитело IgG1κ к CD52)
(9-цис-ретиноевая кислота)
(1,5-дигидро-4H-пиразоло[3,4-d]пиримидин-4-она мононатриевая соль)
(N,N,N',N',N",N"-гексаметил-1,3,5-триазин-2,4,6-триамин)
(этантиол, 2-[(3-аминопропил)амино]-дигидрофосфат (сложный эфир))
(a,a,a',a'-тетраметил-5-(1H-1,2,4-триазол-1-илметил-1,3-бензолдиацетонитрил))
(L-аспарагин-амидогидролаза, тип EC-2)
(лиофилизированный препарат аттенуированного штамма Mycobacterium bovis (Bacillus Calmette-Gukin [BCG], субштамм Montreal)
(4-[1-(5,6,7,8-тетрагидро-3,5,5,8,8-пентаметил-2-нафталенил)-этенил]-бензойная кислота)
(цитотоксические гликопептидные антибиотики, продуцируемые Streptomyces verticillus; блеомицин A2 и блеомицин B2)
(5'-дезокси-5-фтор-N-[(пентилокси)-карбонил]-цитидин)
(диамин-[1,1-циклобутандикарбоксилат(2-)-0,0']-платина,(SP-4-2))
(1,3-бис(2-хлорэтил)-1-нитрозомочевина)
(4-[5-(4-метилфенил)-3-(трифторметил)-1H-пиразол-1-ил]-бензолсульфонамид)
(4-[бис-(2-хлорэтил)-амино]-бензолбутановая кислота)
(PtCl2H6N2)
(2-хлор-2'-дезокси-b-D-аденозин)
(2-[бис(2-хлорэтил)амино]-тетрагидро-2H-13,2-оксазафосфорин-2-оксид моногидрат)
(1-b-D-арабинофуранозилцитозин, C9H13N3O5)
(5-(3,3-диметил-1-триазено)-имидазол-4-карбоксамид (DTIC))
(актиномицин, продуцируемый Streptomyces parvullus, C62H86N12O16)
(рекомбинантный пептид)
((8S-цис)-8-ацетил-10-[(3-амино-2,3,6-тридезокси-á-L-ликсогексопиранозил)окси]-7,8,9,10-тетрагидро-6,8,11-тригидрокси-1-метокси-5,12-нафтацендиона гидрохлорид)
((1S,3S)-3-ацетил-1,2,3,4,6,11-гексагидро-3,5,12-тригидрокси-10-метокси-6,11-диоксо-1-нафтаценил-3-амино-2,3,6-тридезокси-(альфа)-L-ликсогексопиранозида гидрохлорид)
(рекомбинантный пептид)
((S)-4,4'-(1-метил-1,2-этандиил)бис-2,6-пиперазиндион)
((2R,3S)-N-карбокси-3-фенилизосерин, N-трет- бутиловый эфир, 13-эфир с 5b-20-эпокси-12a,4,7b,10b,13a-гексагидрокситакс-11-ен-9-он-4-ацетат-2-бензоатом, тригидрат)
(8S,10S)-10-[(3-амино-2,3,6-тридезокси-a-L-ликсогексопиранозил)окси]-8-гликолил-7,8,9,10-тетрагидро-6,8,11-тригидрокси-1-метокси-5,12-нафтацендиона гидрохлорид)
(17b-гидрокси-2a-метил-5a-андростан-3-она пропионат)
((8S-цис)-10-[(3-амино-2,3,6-тридезокси-a-L-арабино- гексопиранозил)окси]-7,8,9,10-тетрагидро-6,8,11-тригидрокси-8-(гидроксиацетил)-1-метокси-5,12-нафтацендиона гидрохлорид)
(рекомбинантный пептид)
(эстра-1,3,5(10)-триен-3,17-диол(17(бета))-3-[бис(2-хлорэтил)карбамат]-17-(дигидрофосфат), динатриевая соль, моногидрат, или эстрадиол-3-[бис(2-хлорэтил)карбамат]-17-(дигидрофосфат), динатриевая соль, моногидрат)
(4'-деметилэпиподофиллотоксин-9-[4,6-O-(R)-этилиден-(бета)-D-глюкопиранозид], 4'-(дигидрофосфат))
(4'-деметилэпиподофиллотоксин-9-[4,6-0-(R)-этилиден-(бета)-D-глюкопиранозид])
(6-метиленандроста-1,4-диен-3,17-дион)
(рекомбинантный метионил-Г-КСФ человека, r-metHuG-CSF)
(2'-дезокси-5-фторуридин)
(фторированный нуклеотидный аналог противовирусного агента видарабина, 9-b-D-арабинофуранозиладенин (ara-A))
(5-фтор-2,4(1H,3H)-пиримидиндион)
(7-альфа-[9-(4,4,5,5,5-пентафторпентилсульфинил)-нонил]-эстра-1,3,5-(10)-триен-3,17-бета-диол)
(2'-дезокси-2',2'-дифторцитидина моногидрохлорид (b-изомер))
(hP67.6 к CD33)
(иммуноконъюгат, полученный путем образованние ковалентной связи тиомочевины между моноклональным антителом ибритумомабом и линкером-хелатором тиуксетаном [N-[2-бис(карбоксиметил)амино]-3-(п-изотиоцианатофенил)-пропил]-[N-[2-бис(карбоксиметил)амино]-2-(метил)-этил]глицином)
((7S-цис)-9-ацетил-7-[(3-амино-2,3,6-тридезокси-(альфа)-L-ликсо-гексопиранозил)окси]-7,8,9,10-тетрагидро-6,9,11-тригидрокси-5,12-нафтацендиона гидрохлорид)
(3-(2-хлорэтил)-2-[(2-хлорэтил)амино]тетрагидро-2H-1,3,2-оксазафосфорин-2-оксид)
(4-[(4-метил-1-пиперазинил)метил]-N-[4-метил-3-[[4-(3-пиридинил)-2-пиримидинил]амино]-фенил]-бензамида метансульфонат)
(рекомбинантный пептид)
(рекомбинантный пептид)
((4S)-4,11-диэтил-4-гидрокси-9-[(4-пиперидинoпиперидинo)карбонилокси]-1H-пиранo[3',4': 6,7]-индолизино[1,2-b]-хинолин-3,14(4H,12H)-диона гидрохлорид тригидрат)
(4,4'-(1H-1,2,4 -триазол-1-илметилен)-дибензонитрил)
(N-[4-[[(2-амино-5-формил-1,4,5,6,7,8-гексагидро-4-оксо-6-птеридинил)метил]амино]бензоил]-L-глутаминовая кислота, кальциевая соль (1:1))
((-)-(S)-2,3,5,6-тетрагидро-6-фенилимидазо[2,1-b]тиазола моногидрохлорид C11H12N2S·HCl)
(1-(2-хлорэтил)-3-циклогексил-1-нитрозомочевина)
(2-хлор-N-(2-хлорэтил)-N-метилэтанамина гидрохлорид)
17α(ацетилокси)-6-метилпрегна-4,6-диен-3,20-дион
(4-[бис(2-хлорэтил)амино]-L-фенилаланин)
(1,7-дигидро-6H-пурин-6-тиона моногидрат)
(натрия 2-меркаптоэтансульфонат)
(N-[4-[[(2,4-диамино-6-птеридинил)метил]метиламино]бензоил]-L-глутаминовая кислота)
(9-метокси-7H-фуро[3.2-g][1]-бензопиран-7-он)
(1,1-дихлор-2-(o-хлорфенил)-2-(п-хлорфенил)-этан)
(1,4-дигидрокси-5,8-бис[[2-[(2-гидроксиэтил)амино]этил]амино]-9,10-антрацендиона дигидрохлорид)
(IL-11)
(цис-[(1R,2R)-1,2-циклогександиамин-N,N’][оксалато(2-)-O,O’] платина)
(5β,20-эпокси-1,2a,4,7β,10β,13a-гексагидрокситакс-11-ен-9-он-4,10-диацетат-2-бензоат-13-сложный эфир с (2R,3S)-N-бензоил-3-фенилизосерином)
((3-амино-1-гидроксипропилиден)-бис-фосфоновая кислота, динатриевая соль, пентагидрат (APD))
((монометоксиполиэтиленгликоль-сукцинимидил)-11-17-аденозиндезаминаза)
(монометоксиполиэтиленгликоль-укцинимидил-L-аспарагиназа)
(ковалентный конъюгат рекомбинантного метионил-Г-КСФ человека (филграстима) и монометоксиполиэтиленгликоля)
(антибиотик, продуцируемый Streptomyces plicatus)
Canada
(N-изопропил-м-(2-метилгидразино)-п-толуамида моногидрохлорид)
(6-хлор-9-(1-метил-4-диэтиламин)-бутиламино-2-метоксиакридин)
(рекомбинантный пептид)
(рекомбинантное антитело к CD20)
(рекомбинантный пептид)
(стрептозоцин 2-дезокси-2-[[(метилнитрозоамино)-карбонил]-амино]-a(и b)-D-глюкопираноза и 220 мг безводной лимонной кислоты)
(Mg3Si4O10 (OH)2)
((Z)2-[4-(1,2-дифенил-1-бутенил)-фенокси]-N,N-диметилэтанамина 2-гидрокси-1,2,3-пропантрикарбоксилат (1:1))
(3,4-дигидро-3-метил-4-оксоимидазо[5,1-d]-1,2,3,5-тетразин-8-карбоксамид)
(4'-деметилэпиподофиллотоксин 9-[4,6-0-(R)-2-тенилиден-(бета)-D-глюкопиранозид])
(13-гидрокси-3-оксо-13,17-секоандроста-1,4-диен-17-оевой кислоты [dgr]-лактон)
(2-амино-1,7-дигидро-6H-пурин-6-тион)
(1,1',1"-фосфинтиоилидинтрисазиридин, или трис-(1-азиридинил)-фосфинсульфид)
((S)-10-[(диметиламино)метил]-4-этил-4,9-дигидрокси-1H-пиранo[3',4':6,7]индолизино[1,2-b]хинолин-3,14-(4H,12H)-диона моногидрохлорид)
(2-(п-[(Z)-4-хлор-1,2-дифенил-1-бутенил]-фенокси)-N,N-диметилэтиламина цитрат (1:1))
(рекомбинантное иммунотерапевтическое моноклональное антитело мыши IgG2a лямбда к CD20 (с I131 представляет собой радиоиммунотерапевтическое антитело))
(рекомбинантное моноклональное антитело IgG1 каппа к HER2)
(полностью транс-ретиноевая кислота)
((2S-цис)-2-[1,2,3,4,6,11-гексагидро-2,5,12-тригидрокси-7-метокси-6,11-диоксо-4-[[2,3,6-тридезокси-3-[(трифторацетил)-амино-α-L-ликсогексопиранозил]оксил]-2-нафтаценил]-2-оксоэтилпентаноат)
(C46H56N4O10•H2SO4)
(C46H56N4O10•H2SO4)
(3',4'-дидегидро-4'-дезокси-C'-норвинкалейкобластина [R-(R*,R*)-2,3-дигидроксибутандиоат (1:2)(соль)])
((1-гидрокси-2-имидазол-1-ил-фосфонoэтил)-фосфоновая кислота моногидрат)
Противораковые агенты дополнительно включают соединения, которые, как было установлено, обладают противораковой активностью. Примеры включают, но не ограничиваются указанными, 3-AP, 12-O-тетрадеканоилфорбол-13-ацетат, 17AAG, 852A, ABI-007, ABR-217620, ABT-751, ADI-PEG 20, AE-941, AG-013736, AGRO100, аланозин, AMG 706, антитело G250, антинеопластоны, AP23573, апазиквон, APC8015, атипримод, ATN-161, атрасентан, азацитидин, BB-10901, BCX-1777, бевацизумаб, BG00001, бикалутамид, BMS 247550, бортезомиб, бриостатин-1, бусерелин, кальцитриол, CCI-779, CDB-2914, цефиксим, цетуксимаб, CG0070, циленгитид, клофарабин, комбретастатин A4 фосфат, CP-675206, CP-724714, CpG 7909, куркумин, децитабин, DENSPM, доксеркальциферол, E7070, E7389, эктеинасцидин 743, эфапроксирал, эфлорнитин, EKB-569, энзастаурин, эрлотиниб, экзисулинд, фенретинид, флавопиридол, флударабин, флутамид, фотемустин, FR901228, G17DT, галиксимаб, гефитиниб, генистеин, глюфосфамид, GTI-2040, гистрелин, HKI-272, гомогаррингтонин, HSPPC-96, hu14.18-интерлейкин-2 гибридный белок, HuMax-CD4, илопрост, имиквимод, инфликсимаб, интерлейкин-12, IPI-504, ирофульвен, иксабепилон, лапатиниб, леналидомид, лестауртиниб, лейпорид, иммунотоксин LMB-9, лонафарниб, лумиликсимаб, мафосфамид, MB07133, MDX-010, MLN2704, моноклональное антитело 3F8, моноклональное антитело J591, мотексафин, MS-275, MVA-MUC1-IL2, нилутамид, нитрокамптотецин, нолатрекседа дигидрохлорид, нолвадекс, NS-9, O6-бензилгуанин, облимерсен натрия, ONYX-015, ореговомаб, OSI-774, панитумумаб, параплатин, PD-0325901, пеметрексед, PHY906, пиоглитазон, пирфенидон, пиксантрон, PS-341, PSC 833, PXD101, пиразолоакридин, R115777, RAD001, ранпирназа, аналог ребеккамицина, рекомбинантный белок - ангиостатин человека (rhuAngiostatin protein), rhuMab 2C4, розиглитазон, рубитекан, S-1, S-8184, сатраплатин, SB-15992, SGN-0010, SGN-40, сорафениб, SR31747A, ST1571, SU011248, субероиланилидгидроксамовая кислота, сурамин, талабостат, талампанел, тариквидар, темсиролимус, иммунотоксин TGFa-PE38, талидомид, тимальфазин, типифарниб, тирапазамин, TLK286, трабектедин, триметрексата глюкуронат, TroVax, UCN-1, вальпроевая кислота, винфлунин, VNP40101M, волоциксимаб, вориностат, VX-680, ZD1839, ZD6474, зилеутон и зосуквидара тригидрохлорид.
Для более подробного описания противораковых агентов и других терапевтических агентов специалисты в данной области техники могут обратиться к большому количеству инструктивных руководств, включая, но не ограничиваясь ими, источники Physician's Desk Reference (справочник лекарственных средств) и Goodman and Gilman's "Pharmaceutical Basis of Therapeutics" десятое издание, Eds. Hardman et al., 2002.
В некоторых вариантах реализации настоящего изобретения гибридный белок согласно настоящему изобретению и один или более терапевтических агентов или противораковых агентов вводят животному при одном или более из следующих условий: с различной периодичностью, с различной продолжительностью, в различных концентрациях, с помощью различных путей введения и так далее.
Гибридные белки в пределах объема настоящего изобретения включают все гибридные белки, описанные в настоящем документе, причем гибридные белки согласно настоящему изобретению содержатся в количестве, являющемся эффективным для достижения предполагаемой цели настоящего изобретения. Несмотря на то, что индивидуальные потребности различны, определение оптимальных диапазонов эффективных количеств каждого компонента находится в пределах компетенции специалиста в данной области техники. Как правило, соединения могут быть введены млекопитающим, например, людям, перорально в дозе от 0,0025 до 50 мг, или в эквивалентном количестве их фармацевтически приемлемой соли, в день на килограмм массы тела млекопитающего, подлежащего лечению от расстройств, отвечающих на индукцию апоптоза. В одном из вариантов реализации для лечения, облегчения или предотвращения таких расстройств перорально вводят от примерно 0,01 до примерно 25 мг/кг. В случае внутримышечной инъекции доза обычно составляет примерно половину от дозы для перорального введения. Например, подходящая доза для внутримышечного введения будет составлять от примерно 0,0025 до примерно 25 мг/кг или от примерно 0,01 до примерно 5 мг/кг.
Единичная доза для перорального введения может содержать от примерно 0,01 до примерно 1000 мг, например, от примерно 0,1 до примерно 100 мг соединения. Единичная доза может быть введена один или более раз в день в виде одной или более таблеток или капсул, каждая из которых содержит от примерно 0,1 до примерно 10 мг, в целях удобства от примерно 0,25 до 50 мг соединения или его сольватов.
В составе для местного применения соединение может присутствовать в концентрации от примерно 0,01 до 100 мг на грамм носителя. В одном из вариантов реализации соединение присутствует в концентрации примерно 0,07-1,0 мг/мл, например, примерно 0,1-0,5 мг/мл, и в одном из вариантов реализации в концентрации примерно 0,4 мг/мл.
Фармацевтические композиции согласно настоящему изобретению могут быть введены любому пациенту, который может испытывать благоприятное воздействие соединений согласно настоящему изобретению. В первую очередь к таким пациентам относятся млекопитающие, например, люди, хотя не подразумевается, что настоящее изобретение ограничивается этим. Другие пациенты включают подлежащих ветеринарному лечению животных (коров, овец, свиней, лошадей, собак, кошек и тому подобное).
Соединения и их фармацевтические композиции могут быть введены любыми способами, которые обеспечивают достижение цели их применения. Например, введение может осуществляться парентеральным, подкожным, внутривенным, внутримышечным, внутрибрюшинным, чрескожным, буккальным, интратекальным, интракраниальным, интраназальным или местным путями. В качестве альтернативы или одновременно с этим введение может осуществляться пероральным путем. Вводимая доза будет зависеть от возраста, состояния здоровья и массы тела получающего лечение пациента, вида сопутствующего лечения, если таковое имеется, частоты введения и характера желаемого эффекта.
Фармацевтические препараты согласно настоящему изобретению получают известным способом, например, с помощью общепринятых способов смешивания, гранулирования, получения драже, растворения или лиофилизации. Таким образом, фармацевтические препараты для перорального применения могут быть получены путем объединения активных соединений с твердыми вспомогательными веществами, необязательно измельчения полученной смеси и обработки полученной смеси гранул после добавления подходящих вспомогательных веществ, если это желательно или необходимо, с получением таблеток или ядер драже.
Подходящими вспомогательными веществами являются, в частности, наполнители, такие как сахариды, например, лактоза или сахароза, маннитол или сорбитол, препараты целлюлозы и/или фосфаты кальция, например, трикальцийфосфат или гидрофосфат кальция, а также связующие вещества, такие как крахмальная паста, с применением, например, кукурузного крахмала, пшеничного крахмала, рисового крахмала, картофельного крахмала, желатина, трагаканта, метилцеллюлозы, гидроксипропилметилцеллюлозы, натрий-карбоксиметилцеллюлозы и/или поливинилпирролидона. Если желательно, могут быть добавлены разрыхляющие агенты, такие как указанные выше крахмалы, а также карбоксиметилкрахмал, сшитый поливинилпирролидон, агар или альгиновая кислота или ее соль, такая как альгинат натрия. Вспомогательные веществами являются, в первую очередь, регулирующие сыпучесть агенты и смазывающее вещества, например, диоксид кремния, тальк, стеариновая кислота или ее соль, такая как стеарат магния или стеарат кальция, и/или полиэтиленгликоль. Ядра драже обеспечивают подходящими покрытиями, которые, если желательно, являются устойчивыми к действию желудочного сока. Для этой цели могут быть применены концентрированные растворы сахаридов, которые могут необязательно содержать аравийскую камедь, тальк, поливинилпирролидон, полиэтиленгликоль и/или диоксид титана, растворы лакирующих агентов и подходящие органические растворители или смеси растворителей. Для получения покрытий, устойчивых к действию желудочного сока, применяют растворы подходящих препаратов целлюлозы, таких как фталат ацетилцеллюлозы или фталат гидроксипропилметилцеллюлозы. К покрытиям таблеток или драже могут быть добавлены красящие вещества или пигменты, например, для идентификации или для обозначения комбинаций доз активного соединения.
Другие фармацевтические препараты, которые могут быть применены перорально, включают твердые капсулы, полученные из желатина, а также мягкие запаянные капсулы, полученные из желатина и пластификатора, такого как глицерин или сорбитол. Твердые капсулы могут содержать активные соединения в форме гранул, которые могут быть смешаны с наполнителями, такими как лактоза, связующими веществами, такими как крахмалы, и/или смазывающими веществами, такими как тальк или стеарат магния, и, необязательно, стабилизаторами. В мягких капсулах в одном из вариантов реализации активные соединения растворены или суспендированы в подходящих жидкостях, таких как жирные масла или жидкий парафин. Помимо этого, могут быть добавлены стабилизаторы.
Возможные фармацевтические препараты, которые могут быть применены ректально, включают, например, суппозитории, которые состоят из комбинации одного или более активных соединений с суппозиторной основой. Подходящими суппозиторными основами являются, например, природные или синтетические триглицериды или углеводороды парафинового ряда. Помимо этого, также возможно применение желатиновых ректальных капсул, которые состоят из комбинации активных соединений с основой. Возможные вещества, составляющие основу, включают, например, жидкие триглицериды, полиэтиленгликоли или углеводороды парафинового ряда.
Подходящие составы для парентерального введения включают водные растворы активных соединений в водорастворимой форме, например, в форме водорастворимых солей и щелочных растворов. Помимо этого, могут быть введены суспензии активных соединений в виде подходящих масляных суспензий для инъекций. Подходящие липофильные растворители или переносящие среды включают жирные масла, например, кунжутное масло, или синтетические сложные эфиры жирных кислот, например, этилолеат, или триглицериды, или полиэтиленгликоль-400. Водные суспензии для инъекций могут содержать вещества, повышающие вязкость суспензии, включая, например, натрий-карбоксиметилцеллюлозу, сорбитол и/или декстран. Суспензия также может необязательно содержать стабилизаторы.
Композиции для местного применения согласно настоящему изобретению в одном из вариантов реализации приготовлены в виде масел, кремов, лосьонов, мазей и тому подобного путем выбора подходящих носителей. Подходящие носители включают растительные или минеральные масла, белый вазелин (белый мягкий парафин), жиры или масла с разветвленной цепью, животные жиры и высокомолекулярный спирт (более C12). Носители могут быть такими, в которых активный ингредиент растворим. Если желательно, также могут быть включены эмульгирующие агенты, стабилизаторы, смачивающие агенты и антиоксиданты, а также агенты, обеспечивающие цвет или аромат. Помимо этого, в этих составах для местного применения могут быть применены усилители трансдермального проникновения. Примеры таких усилителей можно найти в патентах США № 3989816 и 4444762; каждый из которых полностью включен в настоящий документ посредством ссылки.
Мази могут быть приготовлены путем смешивания раствора активного ингредиента в растительном масле, таком как миндальное масло, с теплым мягким парафином и оставления полученной смеси для остывания. Типичным примером такой мази является мазь, которая содержит примерно 30% миндального масла и примерно 70% белого мягкого парафина по массе. Лосьоны могут быть с легкостью приготовлены путем растворения активного ингредиента в подходящем высокомолекулярном спирте, таком как пропиленгликоль или полиэтиленгликоль.
Специалисту в данной области техники будет легко понять, что изложенное выше представляет собой лишь подробное описание некоторых предпочтительных вариантов реализации настоящего изобретения. Различные модификации и изменения композиций и способов, описанных выше, могут быть легко выполнены с помощью профессиональных знаний и опыта в данной области техники и находятся в пределах объема настоящего изобретения.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Пример 1
Экспрессионные конструкции
В этом примере описаны образцы экспрессионных конструкций, которые вставляют в векторы экспрессии для получения гибридных белков рекомбинантным способом.
Молекула, связывающая VEGF
Последовательность гена для тяжелой цепи гибридного белка бевацизумаб-галакорин (SEQ ID NO:1):
ATGATGTCCTTTGTCTCTCTGCTCCTGGTTGGCATCCTaTTCCATGCCACCCAGGCC GAGGTGCAGCTGGTGGAGTCTGGGGGAGGCTTGGTACAGCCTGGGGGGTCCCTGAGACTCTCCTGTGCAGCCTCTGGATACACCTTTACCAACTATGGCATGAACTGGGTCCGCCAGGCTCCAGGgAAGGGGCTGGAGTGGGTGGGCTGGATAAACACTTACACTGGTGAGCCAACATATGCAGCTGACTTCAAGCGCCGGTTTACCTTCTCTTTGGACACCTCCAAGTCCACGGCCTATCTGCAAATGAACAGCCTGCGGGCCGAGGACACGGCCGTATATTACTGTGCGAAATACCCCCACTACTACGGTAGTAGCCACTGGTACTTTGACGTGTGGGGCCAGGGAACCCTGGTCACCGTCTCCTCAGCCTCCACCAAGGGCCCATCGGTCTTCCCCCTGGCACCCTCCTCCAAGAGCACCTCTGGGGGCACAGCGGCCCTGGGCTGCCTGGTCAAGGACTACTTCCCCGAACCGGTGACGGTGTCGTGGAACTCAGGCGCCCTGACCAGCGGCGTGCACACCTTCCCGGCTGTCCTACAGTCCTCAGGACTCTACTCCCTCAGCAGCGTGGTGACCGTGCCCTCCAGCAGCTTGGGCACCCAGACCTACATCTGCAACGTGAATCACAAGCCCAGCAACACCAAGGTGGACAAGAAAGTTGAGCCCAAATCTTGTGACAAAACTCACACATGCCCACCGTGCCCAGCACCTGAACTCCTGGGGGGACCGTCAGTCTTCCTCTTCCCCCCAAAACCCAAGGACACCCTCATGATCTCCCGGACCCCTGAGGTCACATGCGTGGTGGTGGACGTGAGCCACGAAGACCCTGAGGTCAAGTTCAACTGGTACGTGGACGGCGTGGAGGTGCATAATGCCAAGACAAAGCCGCGGGAGGAGCAGTACAACAGCACGTACCGGGTGGTCAGCGTCCTCACCGTCCTGCACCAGGACTGGCTGAATGGCAAGGAGTACAAGTGCAAGGTCTCCAACAAAGCCCTCCCAGCCCCCATCGAGAAAACCATCTCCAAAGCCAAAGGGCAGCCCCGAGAACCACAGGTGTACACCCTGCCCCCATCCCGGGAGGAGATGACCAAGAACCAGGTCAGCCTGACCTGCCTGGTCAAAGGCTTCTATCCCAGCGACATCGCCGTGGAGTGGGAGAGCAATGGGCAGCCGGAGAACAACTACAAGACCACGCCTCCCGTGCTGGACTCCGACGGCTCCTTCTTCCTCTACAGCAAGCTCACCGTGGACAAGAGCAGGTGGCAGCAGGGGAACGTCTTCTCATGCTCCGTGATGCATGAGGCTCTGCACAACCACTACACGCAGAAGAGCCTCTCCCTGTCTCCCGGGAAA tccgggggtggcggatcc GATGAGGCTGCAGGGATAGGCCCAGAAGTTCCTGATGACCGCGACTTCGAGCCCTCCCTAGGCCCAGTGTGCCCCTTCCGCTGTCAATGCCATCTTCGAGTGGTCCAGTGTTCTGATTTGGGTCTGGACAAAGTGCCAAAGGATCTTCCCCCTGACACAACTCTGCTAGACCTGCAAAACAACAAAATAACCGAAATCAAAGATGGAGACTTTAAGAACCTGAAGAACCTTCACGCATTGATTCTTGTCAACAATAAAATTAGCAAAGTTAGTCCTGGAGCATTTACACCTTTGGTGAAGTTGGAACGACTTTATCTGTCCAAGAATCAGCTGAAGGAATTGCCAGAAAAAATGCCCAAAACTCTTCAGGAGCTGCGTGCCCATGAGAATGAGATCACCAAAGTGCGAAAAGTTACTTTCAATGGACTGAACCAGATGATTGTCATAGAACTGGGCACCAATCCGCTGAAGAGCTCAGGAATTGAAAATGGGGCTTTCCAGGGAATGAAGAAGCTCTCCTACATCCGCATTGCTGATACCAATATCACCAGCATTCCTCAAGGTCTTCCTCCTTCCCTTACGGAATTACATCTTGATGGCAACAAAATCAGCAGAGTTGATGCAGCTAGCCTGAAAGGACTGAATAATTTGGCTAAGTTGGGATTGAGTTTCAACAGCATCTCTGCTGTTGACAATGGCTCTCTGGCCAACACGCCTCATCTGAGGGAGCTTCACTTGGACAACAACAAGCTTACCAGAGTACCTGGTGGGCTGGCAGAGCATAAGTACATCCAGGTTGTCTACCTTCATAACAACAATATCTCTGTAGTTGGATCAAGTGACTTCTGCCCACCTGGACACAACACCAAAAAGGCTTCTTATTCGGGTGTGAGTCTTTTCAGCAACCCGGTCCAGTACTGGGAGATACAGCCATCCACCTTCAGATGTGTCTACGTGCGCTCTGCCATTCAACTCGGAAACTATAAGTGA
Кодирующая сигнальный пептид последовательность выделена жирным шрифтом. Кодирующая тяжелую цепь последовательность подчеркнута прямой линией. Кодирующая линкер последовательность выделена курсивом. Кодирующая галакорин последовательность подчеркнута волнистой линией.
Последовательность белка для тяжелой цепи гибридного белка бевацизумаб-галакорин (SEQ ID NO:2):
MMSFVSLLLVGILFHATQA EVQLVESGGGLVQPGGSLRLSCAASGYTFTNYGMNWVRQAPGKGLEWVGWINTYTGEPTYAADFKRRFTFSLDTSKSTAYLQMNSLRAEDTAVYYCAKYPHYYGSSHWYFDVWGQGTLVTVSSASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKKVEPKSCDKTHTCPPCPAPELLGGPSVFLFPPKPKDTLMISRTPEVTCVVVDVSHEDPEVKFNWYVDGVEVHNAKTKPREEQYNSTYRVVSVLTVLHQDWLNGKEYKCKVSNKALPAPIEKTISKAKGQPREPQVYTLPPSREEMTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSKLTVDKSRWQQGNVFSCSVMHEALHNHYTQKSLSLSPGK SGGGGS DEAAGIGPEVPDDRDFEPSLGPVCPFRCQCHLRVVQCSDLGLDKVPKDLPPDTTLLDLQNNKITEIKDGDFKNLKNLHALILVNNKISKVSPGAFTPLVKLERLYLSKNQLKELPEKMPKTLQELRAHENEITKVRKVTFNGLNQMIVIELGTNPLKSSGIENGAFQGMKKLSYIRIADTNITSIPQGLPPSLTELHLDGNKISRVDAASLKGLNNLAKLGLSFNSISAVDNGSLANTPHLRELHLDNNKLTRVPGGLAEHKYIQVVYLHNNNISVVGSSDFCPPGHNTKKASYSGVSLFSNPVQYWEIQPSTFRCVYVRSAIQLGNYK
Последовательность сигнального пептида выделена жирным шрифтом. Последовательность тяжелой цепи подчеркнута прямой линией. Последовательность линкера выделена курсивом. Последовательность галакорина подчеркнута волнистой линией.
Последовательность гена для легкой цепи бевацизумаба (SEQ ID NO:3):
atgATGTCCTTTGTCTCTCTGCTCCTGGTTGGCATCCTGTTCCATGCCACCCAGGCC GACATCCAGATGACCCAGTCTCCATCCTCCCTGTCTGCATCTGTAGGAGACAGAGTCACCATCACTTGCAGTGCAAGTCAGGACATTAGCAATTATTTAAACTGGTATCAGCAGAAACCAGGGAAAGCTCCTAAGGTCCTGATCTATTTCACATCCAGTTTGCACTCAGGGGTCCCATCTAGGTTCAGTGGCAGTGGATCTGGGACAGATTTCACTCTCACCATCAGCAGCCTGCAGCCTGAAGATTTTGCAACTTATTACTGCCAACAGTATAGTACCGTGCCTTGGACGTTCGGCCAAGGGACCAAGGTGGAAATCAAACGAACTGTGGCTGCACCATCTGTCTTCATCTTCCCGCCATCTGATGAGCAGCTTAAGTCCGGAACTGCTAGCGTTGTGTGCCTGCTGAATAACTTCTATCCCAGAGAGGCCAAAGTACAGTGGAAGGTGGATAACGCCCTCCAATCGGGTAACTCCCAGGAGAGTGTCACAGAGCAGGACAGCAAGGACAGCACCTACAGCCTCAGCAGCACCCTGACGCTGAGCAAAGCAGACTACGAGAAACACAAAGTCTACGCCTGCGAAGTCACCCATCAGGGCCTGAGCTCGCCCGTCACAAAGAGCTTCAACAGGGGAGAGTGTTAG
Кодирующая сигнальный пептид последовательность выделена жирным шрифтом. Кодирующая легкую цепь последовательность подчеркнута прямой линией.
Последовательность белка для легкой цепи бевацизумаба (SEQ ID NO:4):
MMSFVSLLLVGILFHATQA DIQMTQSPSSLSASVGDRVTITCSASQDISNYLNWYQQKPGKAPKVLIYFTSSLHSGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQYSTVPWTFGQGTKVEIKRTVAAPSVFIFPPSDEQLKSGTASVVCLLNNFYPREAKVQWKVDNALQSGNSQESVTEQDSKDSTYSLSSTLTLSKADYEKHKVYACEVTHQGLSSPVTKSFNRGEC
Последовательность сигнального пептида выделена жирным шрифтом. Последовательность легкой цепи подчеркнута прямой линией.
Входящие в состав аминокислотные последовательности:
Сигнальная последовательность для тяжелой и легкой цепей (SEQ ID NO:5):
MMSFVSLLLVGILFHATQA
Последовательность линкера между декорином и тяжелой цепью (SEQ ID NO:6):
SGGGGS
Декорин (SEQ ID NO:7):
DEAAGIGPEVPDDRDFEPSLGPVCPFRCQCHLRVVQCSDLGLDKVPKDLPPDTTLLDLQNNKITEIKDGDFKNLKNLHALILVNNKISKVSPGAFTPLVKLERLYLSKNQLKELPEKMPKTLQELRAHENEITKVRKVTFNGLNQMIVIELGTNPLKSSGIENGAFQGMKKLSYIRIADTNITSIPQGLPPSLTELHLDGNKISRVDAASLKGLNNLAKLGLSFNSISAVDNGSLANTPHLRELHLDNNKLTRVPGGLAEHKYIQVVYLHNNNISVVGSSDFCPPGHNTKKASYSGVSLFSNPVQYWEIQPSTFRCVYVRSAIQLGNYK
Последовательность белка для тяжелой цепи бевацизумаба (SEQ ID NO:8):
EVQLVESGGGLVQPGGSLRLSCAASGYTFTNYGMNWVRQAPGKGLEWVGWINTYTGEPTYAADFKRRFTFSLDTSKSTAYLQMNSLRAEDTAVYYCAKYPHYYGSSHWYFDVWGQGTLVTVSSASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKKVEPKSCDKTHTCPPCPAPELLGGPSVFLFPPKPKDTLMISRTPEVTCVVVDVSHEDPEVKFNWYVDGVEVHNAKTKPREEQYNSTYRVVSVLTVLHQDWLNGKEYKCKVSNKALPAPIEKTISKAKGQPREPQVYTLPPSREEMTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSKLTVDKSRWQQGNVFSCSVMHEALHNHYTQKSLSLSPGK
Последовательность белка для легкой цепи бевацизумаба (SEQ ID NO:9):
DIQMTQSPSSLSASVGDRVTITCSASQDISNYLNWYQQKPGKAPKVLIYFTSSLHSGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQYSTVPWTFGQGTKVEIKRTVAAPSVFIFPPSDEQLKSGTASVVCLLNNFYPREAKVQWKVDNALQSGNSQESVTEQDSKDSTYSLSSTLTLSKADYEKHKVYACEVTHQGLSSPVTKSFNRGEC
Молекула, связывающая CTLA4
Последовательность гена для тяжелой цепи гибридного белка ипилимумаб-галакорин (SEQ ID NO:10):
ATGATGTCCTTTGTCTCTCTGCTCCTGGTTGGCATCCTATTCCATGCCACCCAGGCC caggtgcagctggtggagtccggcggcggcgtcgtgcagcccggccggtccctgcggctgtcctgcgccgcctccggcttcaccttctcctcctacaccatgcactgggtgcggcaggcccccggcaagggcctggagtgggtgactttcatctcctacgacggcaacaacaagtactacgccgactccgtgaagggccggttcaccatctcccgcgacaactccaagaacaccctgtacctgcagatgaactccctgcgggccgaggacaccgccatctactactgcgcccggaccggctggctgggccccttcgactactggggccagggcaccctggtgaccgtgtcctccGCCTCCACCAAGGGCCCATCGGTCTTCCCCCTGGCACCCTCTAGCAAGAGCACCTCTGGGGGCACAGCGGCCCTGGGCTGCCTGGTCAAGGACTACTTCCCCGAACCGGTGACGGTGTCGTGGAACTCAGGCGCCCTGACCAGCGGCGTGCACACCTTCCCGGCTGTCCTACAGTCCTCAGGACTCTACTCCCTCAGCAGCGTGGTGACCGTGCCCTCCAGCAGCTTGGGCACCCAGACCTACATCTGCAACGTGAATCACAAGCCCAGCAACACCAAGGTGGACAAGcggGTTGAGCCCAAATCTTGTGACAAAACTCACACATGCCCACCGTGCCCAGCACCTGAACTCCTGGGGGGACCGTCAGTCTTCCTCTTCCCCCCAAAACCCAAGGACACCCTCATGATCTCCCGGACCCCTGAGGTCACATGCGTGGTGGTGGACGTGAGCCACGAAGACCCTGAGGTCAAGTTCAACTGGTACGTGGACGGCGTGGAGGTGCATAATGCCAAGACAAAGCCGCGGGAGGAGCAGTACAACAGCACGTACCGTGTGGTCAGCGTCCTCACCGTCCTGCACCAGGACTGGCTGAATGGCAAGGAGTACAAGTGCAAGGTCTCCAACAAAGCCCTCCCAGCCCCCATCGAGAAAACCATCTCCAAAGCCAAAGGGCAGCCCCGAGAACCACAGGTGTACACCCTGCCTCCATCCCGcgatGAGCTGACCAAGAACCAGGTCAGCCTGACCTGCCTGGTCAAAGGCTTCTATCCCAGCGACATCGCCGTGGAGTGGGAGAGCAATGGGCAGCCGGAGAACAACTACAAGACCACGCCTCCCGTGCTGGACTCCGACGGCTCCTTCTTCCTCTATAGCAAGCTCACCGTGGACAAGAGCAGGTGGCAGCAGGGGAACGTCTTCTCATGCTCCGTGATGCATGAGGCTCTGCACAACCACTACACGCAGAAGAGCCTCTCCCTGTCTCCtGGGAAA tccgggggtggcggatcc GATGAGGCTGCAGGGATAGGCCCAGAAGTTCCTGATGACCGCGACTTCGAGCCCTCCCTAGGCCCAGTGTGCCCCTTCCGCTGTCAATGCCATCTTCGAGTGGTCCAGTGTTCTGATTTGGGTCTGGACAAAGTGCCAAAGGATCTTCCCCCTGACACAACTCTGCTAGACCTGCAAAACAACAAAATAACCGAAATCAAAGATGGAGACTTTAAGAACCTGAAGAACCTTCACGCATTGATTCTTGTCAACAATAAAATTAGCAAAGTTAGTCCTGGAGCATTTACACCTTTGGTGAAGTTGGAACGACTTTATCTGTCCAAGAATCAGCTGAAGGAATTGCCAGAAAAAATGCCCAAAACTCTTCAGGAGCTGCGTGCCCATGAGAATGAGATCACCAAAGTGCGAAAAGTTACTTTCAATGGACTGAACCAGATGATTGTCATAGAACTGGGCACCAATCCGCTGAAGAGCTCAGGAATTGAAAATGGGGCTTTCCAGGGAATGAAGAAGCTCTCCTACATCCGCATTGCTGATACCAATATCACCAGCATTCCTCAAGGTCTTCCTCCTTCCCTTACGGAATTACATCTTGATGGCAACAAAATCAGCAGAGTTGATGCAGCTAGCCTGAAAGGACTGAATAATTTGGCTAAGTTGGGATTGAGTTTCAACAGCATCTCTGCTGTTGACAATGGCTCTCTGGCCAACACGCCTCATCTGAGGGAGCTTCACTTGGACAACAACAAGCTTACCAGAGTACCTGGTGGGCTGGCAGAGCATAAGTACATCCAGGTTGTCTACCTTCATAACAACAATATCTCTGTAGTTGGATCAAGTGACTTCTGCCCACCTGGACACAACACCAAAAAGGCTTCTTATTCGGGTGTGAGTCTTTTCAGCAACCCGGTCCAGTACTGGGAGATACAGCCATCCACCTTCAGATGTGTCTACGTGCGCTCTGCCATTCAACTCGGAAACTATAAGTGA
Кодирующая сигнальный пептид последовательность выделена жирным шрифтом. Кодирующая тяжелую цепь последовательность подчеркнута прямой линией. Кодирующая линкер последовательность выделена курсивом. Кодирующая галакорин последовательность подчеркнута волнистой линией.
Последовательность белка для тяжелой цепи гибридного белка ипилимумаб-галакорин (SEQ ID NO:11):
MMSFVSLLLVGILFHATQA QVQLVESGGGVVQPGRSLRLSCAASGFTFSSYTMHWVRQAPGKGLEWVTFISYDGNNKYYADSVKGRFTISRDNSKNTLYLQMNSLRAEDTAIYYCARTGWLGPFDYWGQGTLVTVSSASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKRVEPKSCDKTHTCPPCPAPELLGGPSVFLFPPKPKDTLMISRTPEVTCVVVDVSHEDPEVKFNWYVDGVEVHNAKTKPREEQYNSTYRVVSVLTVLHQDWLNGKEYKCKVSNKALPAPIEKTISKAKGQPREPQVYTLPPSRDELTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSKLTVDKSRWQQGNVFSCSVMHEALHNHYTQKSLSLSPGK SGGGGS DEAAGIGPEVPDDRDFEPSLGPVCPFRCQCHLRVVQCSDLGLDKVPKDLPPDTTLLDLQNNKITEIKDGDFKNLKNLHALILVNNKISKVSPGAFTPLVKLERLYLSKNQLKELPEKMPKTLQELRAHENEITKVRKVTFNGLNQMIVIELGTNPLKSSGIENGAFQGMKKLSYIRIADTNITSIPQGLPPSLTELHLDGNKISRVDAASLKGLNNLAKLGLSFNSISAVDNGSLANTPHLRELHLDNNKLTRVPGGLAEHKYIQVVYLHNNNISVVGSSDFCPPGHNTKKASYSGVSLFSNPVQYWEIQPSTFRCVYVRSAIQLGNYK
Последовательность сигнального пептида выделена жирным шрифтом. Последовательность тяжелой цепи подчеркнута прямой линией. Последовательность линкера выделена курсивом. Последовательность галакорина подчеркнута волнистой линией.
Последовательность гена для легкой цепи ипилимумаба (SEQ ID NO:12):
ATGATGTCCTTTGTCTCTCTGCTCCTGGTTGGCATCCTATTCCATGCCACCCAGGCC gagatcgtgctgacccagtcccccggcaccctgtccctgtcccccggcgagcgggccaccctgtcctgccgggcctcccagtccgtgggctcctcctacctggcctggtaccagcagaagcccggccaggccccccggctgctgatctacggcgccttctcccgcgccaccggcatccccgaccggttctccggctccggctccggcaccgacttcaccctgaccatctcccggctggagcccgaggacttcgccgtgtactactgccagcagtacggctcctccccctggaccttcggccagggcaccaaggtggagatcaagcgaACTGTGGCTGCACCATCTGTCTTCATCTTCCCGCCATCTGATGAGCAGCTTAAGTCCGGAACTGCTAGCGTTGTGTGCCTGCTGAATAACTTCTATCCCAGAGAGGCCAAAGTACAGTGGAAGGTGGATAACGCCCTCCAATCGGGTAACTCCCAGGAGAGTGTCACAGAGCAGGACAGCAAGGACAGCACCTACAGCCTCAGCAGCACCCTGACGCTGAGCAAAGCAGACTACGAGAAACACAAAGTCTACGCCTGCGAAGTCACCCATCAGGGCCTGAGCTCGCCCGTCACAAAGAGCTTCAACAGGGGAGAGTGTTAG
Кодирующая сигнальный пептид последовательность выделена жирным шрифтом. Кодирующая легкую цепь последовательность подчеркнута прямой линией.
Последовательность белка для легкой цепи ипилимумаба (SEQ ID NO:13):
MMSFVSLLLVGILFHATQA EIVLTQSPGTLSLSPGERATLSCRASQSVGSSYLAWYQQKPGQAPRLLIYGAFSRATGIPDRFSGSGSGTDFTLTISRLEPEDFAVYYCQQYGSSPWTFGQGTKVEIKRTVAAPSVFIFPPSDEQLKSGTASVVCLLNNFYPREAKVQWKVDNALQSGNSQESVTEQDSKDSTYSLSSTLTLSKADYEKHKVYACEVTHQGLSSPVTKSFNRGEC
Последовательность сигнального пептида выделена жирным шрифтом. Последовательность легкой цепи подчеркнута прямой линией.
Входящие в состав аминокислотные последовательности:
Сигнальная последовательность для тяжелой и легкой цепей (SEQ ID NO:5)
Последовательность линкера между декорином и тяжелой цепью (SEQ ID NO:6)
Декорин (SEQ ID NO:7)
Последовательность белка для тяжелой цепи ипилимумаба (SEQ ID NO:14):
QVQLVESGGGVVQPGRSLRLSCAASGFTFSSYTMHWVRQAPGKGLEWVTFISYDGNNKYYADSVKGRFTISRDNSKNTLYLQMNSLRAEDTAIYYCARTGWLGPFDYWGQGTLVTVSSASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKRVEPKSCDKTHTCPPCPAPELLGGPSVFLFPPKPKDTLMISRTPEVTCVVVDVSHEDPEVKFNWYVDGVEVHNAKTKPREEQYNSTYRVVSVLTVLHQDWLNGKEYKCKVSNKALPAPIEKTISKAKGQPREPQVYTLPPSRDELTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSKLTVDKSRWQQGNVFSCSVMHEALHNHYTQKSLSLSPGK
Последовательность белка для легкой цепи ипилимумаба (SEQ ID NO:15):
EIVLTQSPGTLSLSPGERATLSCRASQSVGSSYLAWYQQKPGQAPRLLIYGAFSRATGIPDRFSGSGSGTDFTLTISRLEPEDFAVYYCQQYGSSPWTFGQGTKVEIKRTVAAPSVFIFPPSDEQLKSGTASVVCLLNNFYPREAKVQWKVDNALQSGNSQESVTEQDSKDSTYSLSSTLTLSKADYEKHKVYACEVTHQGLSSPVTKSFNRGEC
Молекула, связывающая PD-L1
Последовательность гена для тяжелой цепи гибридного белка атезолизумаб-галакорин (SEQ ID NO:16):
ATGATGTCCTTTGTCTCTCTGCTCCTGGTTGGCATCCTATTCCATGCCACCCAGGCC GAGGTACAACTCGTGGAATCCGGCGGGGGACTCGTCCAACCGGGTGGTAGTCTCAGGTTGAGCTGCGCTGCGAGCGGTTTCACTTTCTCAGACTCATGGATTCATTGGGTGCGCCAAGCACCTGGAAAAGGGTTGGAATGGGTTGCCTGGATCTCTCCTTACGGAGGTTCTACCTACTACGCTGATTCAGTAAAGGGGCGGTTCACAATTTCAGCCGATACTTCCAAAAATACGGCTTACCTGCAAATGAACTCTTTGAGGGCGGAGGATACGGCGGTCTACTACTGTGCACGCAGGCACTGGCCTGGAGGGTTCGATTATTGGGGTCAAGGCACTTTGGTAACCGTATCCTCCGCTTCTACCAAAGGCCCATCAGTATTTCCTTTGGCTCCCAGCTCTAAGTCCACTTCCGGTGGAACTGCCGCACTTGGATGTCTCGTCAAAGACTACTTTCCTGAGCCGGTAACTGTGTCATGGAACTCCGGCGCCCTCACTAGCGGCGTCCATACATTTCCAGCGGTTCTCCAGTCAAGTGGCCTCTACAGCCTGTCCAGTGTAGTTACTGTCCCGTCTTCTAGTCTGGGAACGCAAACATATATTTGCAATGTGAATCATAAGCCTAGTAACACAAAAGTCGATAAAAAAGTGGAGCCGAAAAGTTGTGACAAAACGCATACCTGTCCGCCTTGTCCGGCCCCCGAACTCTTGGGCGGCCCATCAGTCTTTCTCTTCCCGCCCAAACCTAAGGACACGTTGATGATAAGTCGCACGCCCGAGGTTACATGCGTCGTAGTCGATGTCAGCCACGAGGATCCGGAGGTAAAGTTTAACTGGTATGTAGACGGAGTTGAAGTACACAACGCCAAAACTAAACCGAGAGAGGAGCAGTACGCATCAACCTATCGCGTAGTATCTGTATTGACGGTCCTTCACCAAGACTGGCTCAATGGGAAAGAATACAAGTGCAAAGTTTCTAATAAAGCCCTCCCTGCACCAATCGAAAAGACTATTTCAAAAGCCAAAGGACAACCAAGAGAACCACAAGTTTATACATTGCCACCTAGTCGCGAGGAGATGACTAAAAACCAAGTGTCCCTTACTTGTCTCGTAAAGGGTTTCTATCCAAGCGACATAGCAGTTGAGTGGGAAAGTAATGGCCAGCCGGAAAACAACTACAAGACGACCCCCCCGGTTCTCGACTCCGATGGATCATTCTTTTTGTATAGTAAACTCACAGTTGATAAGAGTCGATGGCAGCAGGGGAATGTTTTTTCTTGCTCTGTGATGCACGAGGCGCTCCACAACCACTATACGCAAAAGTCCCTCAGCCTGAGCCCCGGGAAA TCCGGGGGTGGCGGATCC GATGAGGCTGCAGGGATAGGCCCAGAAGTTCCTGATGACCGCGACTTCGAGCCCTCCCTAGGCCCAGTGTGCCCCTTCCGCTGTCAATGCCATCTTCGAGTGGTCCAGTGTTCTGATTTGGGTCTGGACAAAGTGCCAAAGGATCTTCCCCCTGACACAACTCTGCTAGACCTGCAAAACAACAAAATAACCGAAATCAAAGATGGAGACTTTAAGAACCTGAAGAACCTTCACGCATTGATTCTTGTCAACAATAAAATTAGCAAAGTTAGTCCTGGAGCATTTACACCTTTGGTGAAGTTGGAACGACTTTATCTGTCCAAGAATCAGCTGAAGGAATTGCCAGAAAAAATGCCCAAAACTCTTCAGGAGCTGCGTGCCCATGAGAATGAGATCACCAAAGTGCGAAAAGTTACTTTCAATGGACTGAACCAGATGATTGTCATAGAACTGGGCACCAATCCGCTGAAGAGCTCAGGAATTGAAAATGGGGCTTTCCAGGGAATGAAGAAGCTCTCCTACATCCGCATTGCTGATACCAATATCACCAGCATTCCTCAAGGTCTTCCTCCTTCCCTTACGGAATTACATCTTGATGGCAACAAAATCAGCAGAGTTGATGCAGCTAGCCTGAAAGGACTGAATAATTTGGCTAAGTTGGGATTGAGTTTCAACAGCATCTCTGCTGTTGACAATGGCTCTCTGGCCAACACGCCTCATCTGAGGGAGCTTCACTTGGACAACAACAAGCTTACCAGAGTACCTGGTGGGCTGGCAGAGCATAAGTACATCCAGGTTGTCTACCTTCATAACAACAATATCTCTGTAGTTGGATCAAGTGACTTCTGCCCACCTGGACACAACACCAAAAAGGCTTCTTATTCGGGTGTGAGTCTTTTCAGCAACCCGGTCCAGTACTGGGAGATACAGCCATCCACCTTCAGATGTGTCTACGTGCGCTCTGCCATTCAACTCGGAAACTATAAGTGA
Кодирующая сигнальный пептид последовательность выделена жирным шрифтом. Кодирующая тяжелую цепь последовательность подчеркнута прямой линией. Кодирующая линкер последовательность выделена курсивом. Кодирующая галакорин последовательность подчеркнута волнистой линией.
Последовательность белка для тяжелой цепи гибридного белка атезолизумаб-галакорин (SEQ ID NO:17):
MMSFVSLLLVGILFHATQA EVQLVESGGGLVQPGGSLRLSCAASGFTFSDSWIHWVRQAPGKGLEWVAWISPYGGSTYYADSVKGRFTISADTSKNTAYLQMNSLRAEDTAVYYCARRHWPGGFDYWGQGTLVTVSSASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKKVEPKSCDKTHTCPPCPAPELLGGPSVFLFPPKPKDTLMISRTPEVTCVVVDVSHEDPEVKFNWYVDGVEVHNAKTKPREEQYASTYRVVSVLTVLHQDWLNGKEYKCKVSNKALPAPIEKTISKAKGQPREPQVYTLPPSREEMTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSKLTVDKSRWQQGNVFSCSVMHEALHNHYTQKSLSLSPGK SGGGGS DEAAGIGPEVPDDRDFEPSLGPVCPFRCQCHLRVVQCSDLGLDKVPKDLPPDTTLLDLQNNKITEIKDGDFKNLKNLHALILVNNKISKVSPGAFTPLVKLERLYLSKNQLKELPEKMPKTLQELRAHENEITKVRKVTFNGLNQMIVIELGTNPLKSSGIENGAFQGMKKLSYIRIADTNITSIPQGLPPSLTELHLDGNKISRVDAASLKGLNNLAKLGLSFNSISAVDNGSLANTPHLRELHLDNNKLTRVPGGLAEHKYIQVVYLHNNNISVVGSSDFCPPGHNTKKASYSGVSLFSNPVQYWEIQPSTFRCVYVRSAIQLGNYK
Последовательность сигнального пептида выделена жирным шрифтом. Последовательность тяжелой цепи подчеркнута прямой линией. Последовательность линкера выделена курсивом. Последовательность галакорина подчеркнута волнистой линией.
Последовательность гена для легкой цепи атезолизумаба (SEQ ID NO:18):
ATGATGTCCTTTGTCTCTCTGCTCCTGGTTGGCATCCTATTCCATGCCACCCAGGCC GACATTCAGATGACACAATCACCTAGCAGTCTGAGTGCGAGCGTAGGTGATCGCGTAACGATTACCTGCAGGGCCTCTCAAGACGTGTCAACGGCAGTGGCGTGGTACCAGCAGAAGCCTGGTAAAGCTCCTAAGCTCCTCATCTATTCAGCTTCCTTCTTGTATAGTGGAGTACCGTCAAGATTTTCCGGAAGCGGATCAGGTACAGATTTTACTTTGACTATCAGTAGTTTGCAGCCAGAGGATTTCGCTACATATTACTGTCAACAATATCTCTATCACCCTGCCACTTTTGGACAAGGGACTAAAGTCGAAATAAAACGAACAGTGGCCGCACCAAGCGTTTTTATCTTTCCCCCATCCGACGAGCAGTTGAAGAGCGGCACCGCGTCCGTGGTCTGCCTGTTGAATAATTTCTATCCAAGGGAGGCAAAAGTGCAATGGAAAGTTGATAATGCGCTTCAATCCGGAAACTCACAAGAATCAGTAACAGAACAAGACTCTAAAGACAGTACATATTCTCTTAGTAGCACACTCACTCTTTCAAAGGCTGACTATGAGAAACATAAAGTGTACGCTTGTGAAGTGACACATCAAGGTCTTAGCTCCCCAGTAACTAAGAGCTTTAATAGGGGCGAGTGCTGA
Кодирующая сигнальный пептид последовательность выделена жирным шрифтом. Кодирующая легкую цепь последовательность подчеркнута прямой линией.
Последовательность белка для легкой цепи атезолизумаба (SEQ ID NO:19):
MMSFVSLLLVGILFHATQA DIQMTQSPSSLSASVGDRVTITCRASQDVSTAVAWYQQKPGKAPKLLIYSASFLYSGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQYLYHPATFGQGTKVEIKRTVAAPSVFIFPPSDEQLKSGTASVVCLLNNFYPREAKVQWKVDNALQSGNSQESVTEQDSKDSTYSLSSTLTLSKADYEKHKVYACEVTHQGLSSPVTKSFNRGEC
Последовательность сигнального пептида выделена жирным шрифтом. Последовательность легкой цепи подчеркнута прямой линией.
Входящие в состав аминокислотные последовательности:
Сигнальная последовательность для тяжелой и легкой цепей (SEQ ID NO:5)
Последовательность линкера между декорином и тяжелой цепью (SEQ ID NO:6)
Декорин (SEQ ID NO:7)
Последовательность белка для тяжелой цепи атезолизумаба (SEQ ID NO:20):
EVQLVESGGGLVQPGGSLRLSCAASGFTFSDSWIHWVRQAPGKGLEWVAWISPYGGSTYYADSVKGRFTISADTSKNTAYLQMNSLRAEDTAVYYCARRHWPGGFDYWGQGTLVTVSSASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKKVEPKSCDKTHTCPPCPAPELLGGPSVFLFPPKPKDTLMISRTPEVTCVVVDVSHEDPEVKFNWYVDGVEVHNAKTKPREEQYASTYRVVSVLTVLHQDWLNGKEYKCKVSNKALPAPIEKTISKAKGQPREPQVYTLPPSREEMTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSKLTVDKSRWQQGNVFSCSVMHEALHNHYTQKSLSLSPGK
Последовательность белка для легкой цепи атезолизумаба (SEQ ID NO:21):
DIQMTQSPSSLSASVGDRVTITCRASQDVSTAVAWYQQKPGKAPKLLIYSASFLYSGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQYLYHPATFGQGTKVEIKRTVAAPSVFIFPPSDEQLKSGTASVVCLLNNFYPREAKVQWKVDNALQSGNSQESVTEQDSKDSTYSLSSTLTLSKADYEKHKVYACEVTHQGLSSPVTKSFNRGEC
Молекула, связывающая PD-L1
Последовательность гена для тяжелой цепи гибридного белка авелумаб-галакорин (SEQ ID NO:22):
ATGATGTCCTTTGTCTCTCTGCTCCTGGTTGGCATCCTATTCCATGCCACCCAGGCC GAGGTACAGCTTTTGGAGTCAGGCGGGGGGCTCGTCCAACCTGGGGGGTCACTCCGGTTGTCATGTGCTGCCAGTGGCTTCACATTCTCATCTTACATTATGATGTGGGTTCGACAGGCCCCTGGGAAGGGGCTGGAGTGGGTTTCCTCCATCTACCCCTCCGGGGGAATTACCTTCTATGCCGACACGGTAAAGGGTCGCTTCACTATAAGTCGAGATAACAGCAAAAATACGCTGTATCTCCAGATGAACTCTCTCAGGGCTGAGGACACAGCTGTATATTACTGCGCGCGGATTAAGTTGGGGACCGTCACAACAGTGGATTACTGGGGTCAAGGCACTCTGGTAACCGTATCCTCAGCATCCACCAAGGGCCCAAGTGTATTCCCGCTGGCCCCTTCAAGTAAATCCACGTCTGGCGGCACAGCCGCTCTCGGTTGCCTGGTTAAGGACTACTTCCCAGAACCTGTCACTGTCAGTTGGAACTCAGGCGCATTGACATCTGGTGTCCATACATTCCCCGCAGTCCTGCAAAGCTCTGGACTTTACAGTCTTAGTAGCGTAGTGACAGTCCCATCTTCAAGTCTTGGGACCCAAACTTATATTTGCAACGTAAATCATAAACCCTCCAACACTAAAGTAGACAAAAAAGTAGAGCCGAAATCTTGCGACAAAACGCATACATGCCCACCATGTCCCGCTCCGGAACTCCTGGGCGGCCCGTCCGTTTTTCTCTTTCCCCCAAAGCCCAAGGATACGCTTATGATCAGCAGAACACCGGAAGTTACTTGTGTAGTCGTTGACGTGTCTCACGAAGATCCCGAAGTCAAATTTAATTGGTATGTGGATGGCGTCGAAGTGCACAACGCAAAAACCAAACCCAGAGAGGAACAGTATAACAGCACGTATCGAGTGGTCTCCGTACTTACGGTCCTCCACCAGGACTGGTTGAATGGCAAGGAGTACAAGTGCAAAGTGAGCAATAAAGCGTTGCCAGCCCCGATCGAAAAAACCATCAGCAAGGCCAAGGGACAGCCTAGAGAGCCGCAGGTTTACACCTTGCCGCCATCAAGGGATGAACTGACTAAAAACCAGGTATCCCTGACCTGCCTGGTTAAGGGTTTTTACCCCAGTGATATAGCGGTTGAATGGGAGTCTAACGGGCAGCCAGAGAACAACTACAAAACGACACCTCCCGTTCTGGATTCCGATGGCAGCTTTTTCTTGTATTCTAAACTCACCGTGGATAAATCCCGATGGCAGCAAGGCAACGTCTTCTCCTGCAGCGTGATGCATGAAGCCTTGCACAACCACTATACCCAAAAGAGTCTCAGCCTGTCACCcGGGAAA TCCGGGGGTGGCGGATCC GATGAGGCTGCAGGGATAGGCCCAGAAGTTCCTGATGACCGCGACTTCGAGCCCTCCCTAGGCCCAGTGTGCCCCTTCCGCTGTCAATGCCATCTTCGAGTGGTCCAGTGTTCTGATTTGGGTCTGGACAAAGTGCCAAAGGATCTTCCCCCTGACACAACTCTGCTAGACCTGCAAAACAACAAAATAACCGAAATCAAAGATGGAGACTTTAAGAACCTGAAGAACCTTCACGCATTGATTCTTGTCAACAATAAAATTAGCAAAGTTAGTCCTGGAGCATTTACACCTTTGGTGAAGTTGGAACGACTTTATCTGTCCAAGAATCAGCTGAAGGAATTGCCAGAAAAAATGCCCAAAACTCTTCAGGAGCTGCGTGCCCATGAGAATGAGATCACCAAAGTGCGAAAAGTTACTTTCAATGGACTGAACCAGATGATTGTCATAGAACTGGGCACCAATCCGCTGAAGAGCTCAGGAATTGAAAATGGGGCTTTCCAGGGAATGAAGAAGCTCTCCTACATCCGCATTGCTGATACCAATATCACCAGCATTCCTCAAGGTCTTCCTCCTTCCCTTACGGAATTACATCTTGATGGCAACAAAATCAGCAGAGTTGATGCAGCTAGCCTGAAAGGACTGAATAATTTGGCTAAGTTGGGATTGAGTTTCAACAGCATCTCTGCTGTTGACAATGGCTCTCTGGCCAACACGCCTCATCTGAGGGAGCTTCACTTGGACAACAACAAGCTTACCAGAGTACCTGGTGGGCTGGCAGAGCATAAGTACATCCAGGTTGTCTACCTTCATAACAACAATATCTCTGTAGTTGGATCAAGTGACTTCTGCCCACCTGGACACAACACCAAAAAGGCTTCTTATTCGGGTGTGAGTCTTTTCAGCAACCCGGTCCAGTACTGGGAGATACAGCCATCCACCTTCAGATGTGTCTACGTGCGCTCTGCCATTCAACTCGGAAACTATAAGTGA
Кодирующая сигнальный пептид последовательность выделена жирным шрифтом. Кодирующая тяжелую цепь последовательность подчеркнута прямой линией. Кодирующая линкер последовательность выделена курсивом. Кодирующая галакорин последовательность подчеркнута волнистой линией.
Последовательность белка для тяжелой цепи гибридного белка авелумаб-галакорин (SEQ ID NO:23):
MMSFVSLLLVGILFHATQA EVQLLESGGGLVQPGGSLRLSCAASGFTFSSYIMMWVRQAPGKGLEWVSSIYPSGGITFYADTVKGRFTISRDNSKNTLYLQMNSLRAEDTAVYYCARIKLGTVTTVDYWGQGTLVTVSSASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKKVEPKSCDKTHTCPPCPAPELLGGPSVFLFPPKPKDTLMISRTPEVTCVVVDVSHEDPEVKFNWYVDGVEVHNAKTKPREEQYNSTYRVVSVLTVLHQDWLNGKEYKCKVSNKALPAPIEKTISKAKGQPREPQVYTLPPSRDELTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSKLTVDKSRWQQGNVFSCSVMHEALHNHYTQKSLSLSPGK SGGGGS DEAAGIGPEVPDDRDFEPSLGPVCPFRCQCHLRVVQCSDLGLDKVPKDLPPDTTLLDLQNNKITEIKDGDFKNLKNLHALILVNNKISKVSPGAFTPLVKLERLYLSKNQLKELPEKMPKTLQELRAHENEITKVRKVTFNGLNQMIVIELGTNPLKSSGIENGAFQGMKKLSYIRIADTNITSIPQGLPPSLTELHLDGNKISRVDAASLKGLNNLAKLGLSFNSISAVDNGSLANTPHLRELHLDNNKLTRVPGGLAEHKYIQVVYLHNNNISVVGSSDFCPPGHNTKKASYSGVSLFSNPVQYWEIQPSTFRCVYVRSAIQLGNYK
Последовательность сигнального пептида выделена жирным шрифтом. Последовательность тяжелой цепи подчеркнута прямой линией. Последовательность линкера выделена курсивом. Последовательность галакорина подчеркнута волнистой линией.
Последовательность гена для легкой цепи авелумаба (SEQ ID NO:24):
ATGATGTCCTTTGTCTCTCTGCTCCTGGTTGGCATCCTATTCCATGCCACCCAGGCC CAGTCTGCACTTACACAACCGGCGTCCGTTTCCGGATCTCCAGGACAGAGCATTACTATCAGTTGCACGGGAACCTCCTCAGACGTAGGGGGGTATAATTATGTGTCTTGGTATCAACAGCATCCCGGGAAAGCCCCCAAACTGATGATCTACGATGTCAGCAATAGACCAAGCGGTGTGAGTAATCGATTTAGCGGGTCTAAATCTGGTAACACAGCATCCCTCACTATTAGTGGACTGCAAGCAGAAGATGAGGCAGACTATTATTGCAGTAGCTATACGTCTAGTTCCACCCGCGTTTTTGGCACTGGGACGAAAGTCACCGTTCTCGGACAACCAAAAGCAAACCCCACCGTGACTCTGTTTCCGCCTAGCAGCGAAGAATTGCAGGCCAATAAGGCGACACTCGTATGCCTTATCTCCGACTTCTACCCGGGCGCTGTGACAGTCGCGTGGAAAGCCGACGGCAGCCCTGTTAAAGCTGGAGTCGAGACCACGAAGCCGTCCAAGCAGAGTAACAATAAGTATGCTGCATCCAGTTATCTCTCTCTCACTCCGGAACAGTGGAAGTCCCATCGGTCCTATAGTTGCCAAGTGACCCATGAGGGTTCCACCGTAGAGAAAACGGTAGCACCTACCGAATGTAGTTGA
Кодирующая сигнальный пептид последовательность выделена жирным шрифтом. Кодирующая легкую цепь последовательность подчеркнута прямой линией.
Последовательность белка для легкой цепи авелумаба (SEQ ID NO:25):
MMSFVSLLLVGILFHATQA QSALTQPASVSGSPGQSITISCTGTSSDVGGYNYVSWYQQHPGKAPKLMIYDVSNRPSGVSNRFSGSKSGNTASLTISGLQAEDEADYYCSSYTSSSTRVFGTGTKVTVLGQPKANPTVTLFPPSSEELQANKATLVCLISDFYPGAVTVAWKADGSPVKAGVETTKPSKQSNNKYAASSYLSLTPEQWKSHRSYSCQVTHEGSTVEKTVAPTECS
Последовательность сигнального пептида выделена жирным шрифтом. Последовательность легкой цепи подчеркнута прямой линией.
Входящие в состав аминокислотные последовательности:
Сигнальная последовательность для тяжелой и легкой цепей (SEQ ID NO:5)
Последовательность линкера между декорином и тяжелой цепью (SEQ ID NO:6)
Декорин (SEQ ID NO:7)
Последовательность белка для тяжелой цепи авелумаба (SEQ ID NO:26):
EVQLLESGGGLVQPGGSLRLSCAASGFTFSSYIMMWVRQAPGKGLEWVSSIYPSGGITFYADTVKGRFTISRDNSKNTLYLQMNSLRAEDTAVYYCARIKLGTVTTVDYWGQGTLVTVSSASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKKVEPKSCDKTHTCPPCPAPELLGGPSVFLFPPKPKDTLMISRTPEVTCVVVDVSHEDPEVKFNWYVDGVEVHNAKTKPREEQYNSTYRVVSVLTVLHQDWLNGKEYKCKVSNKALPAPIEKTISKAKGQPREPQVYTLPPSRDELTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSKLTVDKSRWQQGNVFSCSVMHEALHNHYTQKSLSLSPGK
Последовательность белка для легкой цепи авелумаба (SEQ ID NO:27):
QSALTQPASVSGSPGQSITISCTGTSSDVGGYNYVSWYQQHPGKAPKLMIYDVSNRPSGVSNRFSGSKSGNTASLTISGLQAEDEADYYCSSYTSSSTRVFGTGTKVTVLGQPKANPTVTLFPPSSEELQANKATLVCLISDFYPGAVTVAWKADGSPVKAGVETTKPSKQSNNKYAASSYLSLTPEQWKSHRSYSCQVTHEGSTVEKTVAPTECS
Молекула, связывающая PD-L1
Последовательность гена для тяжелой цепи гибридного белка дурвалумаб-галакорин (SEQ ID NO:28):
ATGATGTCCTTTGTCTCTCTGCTCCTGGTTGGCATCCTATTCCATGCCACCCAGGCC GAGGTCCAGCTTGTTGAAAGCGGTGGTGGCCTCGTGCAGCCTGGTGGCAGTTTGCGGTTGTCTTGCGCAGCTAGTGGTTTTACCTTCTCCAGATACTGGATGTCATGGGTCCGACAGGCCCCTGGCAAGGGGCTTGAATGGGTTGCAAATATAAAGCAGGACGGTTCTGAAAAGTACTATGTAGACTCCGTCAAAGGAAGATTTACTATTAGTCGAGACAACGCCAAGAATAGCCTCTACCTTCAGATGAATTCTTTGCGAGCGGAGGACACAGCCGTATATTACTGCGCACGAGAAGGGGGGTGGTTCGGTGAACTGGCTTTTGACTACTGGGGGCAAGGTACGCTTGTCACGGTGAGCTCTGCCTCTACAAAGGGGCCGTCTGTGTTTCCACTTGCTCCATCTAGTAAGTCAACTTCTGGAGGTACTGCGGCATTGGGATGCCTTGTTAAGGATTATTTTCCCGAACCTGTAACTGTGAGCTGGAATTCAGGTGCCCTCACCTCTGGTGTACATACCTTTCCAGCAGTTTTGCAATCTTCCGGTTTGTACTCTCTTAGTTCAGTTGTAACTGTCCCCTCTTCCTCTCTTGGTACCCAAACATACATTTGTAATGTCAATCACAAACCAAGCAATACCAAGGTAGACAAGCGGGTGGAACCCAAATCTTGTGACAAAACTCATACCTGCCCACCATGTCCCGCCCCGGAGTTTGAAGGAGGTCCAAGTGTATTTCTTTTCCCGCCTAAGCCTAAGGATACCCTCATGATAAGTCGGACACCAGAGGTGACGTGTGTTGTGGTAGACGTGAGTCACGAAGATCCCGAAGTTAAATTTAATTGGTATGTGGACGGGGTGGAAGTCCATAACGCGAAGACAAAGCCACGCGAAGAGCAGTACAATTCCACGTACAGGGTGGTTAGCGTGCTTACCGTCCTGCATCAAGATTGGCTGAACGGGAAAGAATACAAATGCAAAGTATCCAACAAGGCGTTGCCTGCGAGTATCGAGAAAACGATTTCTAAAGCTAAAGGACAACCCCGGGAACCCCAGGTCTATACACTGCCGCCCAGCAGAGAAGAGATGACGAAAAATCAAGTATCCCTTACGTGTCTCGTCAAAGGCTTCTACCCTTCCGATATTGCTGTAGAGTGGGAATCTAACGGGCAGCCGGAAAATAACTACAAGACTACTCCGCCAGTACTTGATTCAGACGGCTCCTTCTTCCTTTATTCAAAACTCACCGTAGATAAAAGTAGGTGGCAACAAGGTAATGTTTTTAGCTGTAGCGTAATGCACGAAGCGTTGCATAACCATTATACACAGAAATCACTCAGCCTGTCCCCCGGGAAA TCCGGGGGTGGCGGATCC GATGAGGCTGCAGGGATAGGCCCAGAAGTTCCTGATGACCGCGACTTCGAGCCCTCCCTAGGCCCAGTGTGCCCCTTCCGCTGTCAATGCCATCTTCGAGTGGTCCAGTGTTCTGATTTGGGTCTGGACAAAGTGCCAAAGGATCTTCCCCCTGACACAACTCTGCTAGACCTGCAAAACAACAAAATAACCGAAATCAAAGATGGAGACTTTAAGAACCTGAAGAACCTTCACGCATTGATTCTTGTCAACAATAAAATTAGCAAAGTTAGTCCTGGAGCATTTACACCTTTGGTGAAGTTGGAACGACTTTATCTGTCCAAGAATCAGCTGAAGGAATTGCCAGAAAAAATGCCCAAAACTCTTCAGGAGCTGCGTGCCCATGAGAATGAGATCACCAAAGTGCGAAAAGTTACTTTCAATGGACTGAACCAGATGATTGTCATAGAACTGGGCACCAATCCGCTGAAGAGCTCAGGAATTGAAAATGGGGCTTTCCAGGGAATGAAGAAGCTCTCCTACATCCGCATTGCTGATACCAATATCACCAGCATTCCTCAAGGTCTTCCTCCTTCCCTTACGGAATTACATCTTGATGGCAACAAAATCAGCAGAGTTGATGCAGCTAGCCTGAAAGGACTGAATAATTTGGCTAAGTTGGGATTGAGTTTCAACAGCATCTCTGCTGTTGACAATGGCTCTCTGGCCAACACGCCTCATCTGAGGGAGCTTCACTTGGACAACAACAAGCTTACCAGAGTACCTGGTGGGCTGGCAGAGCATAAGTACATCCAGGTTGTCTACCTTCATAACAACAATATCTCTGTAGTTGGATCAAGTGACTTCTGCCCACCTGGACACAACACCAAAAAGGCTTCTTATTCGGGTGTGAGTCTTTTCAGCAACCCGGTCCAGTACTGGGAGATACAGCCATCCACCTTCAGATGTGTCTACGTGCGCTCTGCCATTCAACTCGGAAACTATAAGTGA
Кодирующая сигнальный пептид последовательность выделена жирным шрифтом. Кодирующая тяжелую цепь последовательность подчеркнута прямой линией. Кодирующая линкер последовательность выделена курсивом. Кодирующая галакорин последовательность подчеркнута волнистой линией.
Последовательность белка для тяжелой цепи гибридного белка дурвалумаб-галакорин (SEQ ID NO:29):
MMSFVSLLLVGILFHATQA EVQLVESGGGLVQPGGSLRLSCAASGFTFSRYWMSWVRQAPGKGLEWVANIKQDGSEKYYVDSVKGRFTISRDNAKNSLYLQMNSLRAEDTAVYYCAREGGWFGELAFDYWGQGTLVTVSSASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKRVEPKSCDKTHTCPPCPAPEFEGGPSVFLFPPKPKDTLMISRTPEVTCVVVDVSHEDPEVKFNWYVDGVEVHNAKTKPREEQYNSTYRVVSVLTVLHQDWLNGKEYKCKVSNKALPASIEKTISKAKGQPREPQVYTLPPSREEMTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSKLTVDKSRWQQGNVFSCSVMHEALHNHYTQKSLSLSPGK SGGGGS DEAAGIGPEVPDDRDFEPSLGPVCPFRCQCHLRVVQCSDLGLDKVPKDLPPDTTLLDLQNNKITEIKDGDFKNLKNLHALILVNNKISKVSPGAFTPLVKLERLYLSKNQLKELPEKMPKTLQELRAHENEITKVRKVTFNGLNQMIVIELGTNPLKSSGIENGAFQGMKKLSYIRIADTNITSIPQGLPPSLTELHLDGNKISRVDAASLKGLNNLAKLGLSFNSISAVDNGSLANTPHLRELHLDNNKLTRVPGGLAEHKYIQVVYLHNNNISVVGSSDFCPPGHNTKKASYSGVSLFSNPVQYWEIQPSTFRCVYVRSAIQLGNYK
Последовательность сигнального пептида выделена жирным шрифтом. Последовательность тяжелой цепи подчеркнута прямой линией. Последовательность линкера выделена курсивом. Последовательность галакорина подчеркнута волнистой линией.
Последовательность гена для легкой цепи дурвалумаба (SEQ ID NO:30):
ATGATGTCCTTTGTCTCTCTGCTCCTGGTTGGCATCCTATTCCATGCCACCCAGGCC GAGATAGTTTTGACTCAAAGCCCTGGAACGCTCTCTTTGTCTCCCGGCGAGCGGGCGACCCTTTCCTGTAGGGCTAGCCAGAGGGTATCTTCCTCTTACCTGGCATGGTACCAGCAAAAGCCCGGACAAGCCCCCCGACTTCTGATTTATGACGCCTCATCCCGGGCGACAGGCATCCCTGACCGATTTTCAGGGAGTGGCTCTGGTACCGATTTTACGCTTACGATTTCCAGGCTGGAGCCCGAGGATTTCGCAGTGTATTACTGTCAACAATACGGCAGCTTGCCCTGGACCTTTGGACAAGGAACCAAGGTAGAGATCAAAAGGACCGTTGCCGCCCCGTCCGTGTTCATCTTCCCTCCGAGCGATGAGCAACTTAAAAGTGGAACTGCAAGCGTTGTATGTCTTCTGAACAATTTCTATCCCCGAGAAGCCAAGGTACAGTGGAAAGTGGATAATGCCCTCCAATCTGGCAATAGCCAAGAGTCTGTCACAGAGCAGGACAGCAAGGACTCAACTTATTCACTTAGCTCCACCCTGACGCTGAGTAAAGCAGACTACGAGAAGCATAAGGTGTATGCTTGTGAGGTTACACACCAAGGCTTGTCTTCTCCTGTCACGAAGTCTTTCAATAGGGGCGAATGCTGA
Кодирующая сигнальный пептид последовательность выделена жирным шрифтом. Кодирующая легкую цепь последовательность подчеркнута прямой линией.
Последовательность белка для легкой цепи дурвалумаба (SEQ ID NO:31):
MMSFVSLLLVGILFHATQA EIVLTQSPGTLSLSPGERATLSCRASQRVSSSYLAWYQQKPGQAPRLLIYDASSRATGIPDRFSGSGSGTDFTLTISRLEPEDFAVYYCQQYGSLPWTFGQGTKVEIKRTVAAPSVFIFPPSDEQLKSGTASVVCLLNNFYPREAKVQWKVDNALQSGNSQESVTEQDSKDSTYSLSSTLTLSKADYEKHKVYACEVTHQGLSSPVTKSFNRGEC
Последовательность сигнального пептида выделена жирным шрифтом. Последовательность легкой цепи подчеркнута прямой линией.
Входящие в состав аминокислотные последовательности:
Сигнальная последовательность для тяжелой и легкой цепей (SEQ ID NO:5)
Последовательность линкера между декорином и тяжелой цепью (SEQ ID NO:6)
Декорин (SEQ ID NO:7)
Последовательность белка для тяжелой цепи дурвалумаба (SEQ ID NO:32):
EVQLVESGGGLVQPGGSLRLSCAASGFTFSRYWMSWVRQAPGKGLEWVANIKQDGSEKYYVDSVKGRFTISRDNAKNSLYLQMNSLRAEDTAVYYCAREGGWFGELAFDYWGQGTLVTVSSASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKRVEPKSCDKTHTCPPCPAPEFEGGPSVFLFPPKPKDTLMISRTPEVTCVVVDVSHEDPEVKFNWYVDGVEVHNAKTKPREEQYNSTYRVVSVLTVLHQDWLNGKEYKCKVSNKALPASIEKTISKAKGQPREPQVYTLPPSREEMTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSKLTVDKSRWQQGNVFSCSVMHEALHNHYTQKSLSLSPGK
Последовательность белка для легкой цепи дурвалумаба (SEQ ID NO:33):
EIVLTQSPGTLSLSPGERATLSCRASQRVSSSYLAWYQQKPGQAPRLLIYDASSRATGIPDRFSGSGSGTDFTLTISRLEPEDFAVYYCQQYGSLPWTFGQGTKVEIKRTVAAPSVFIFPPSDEQLKSGTASVVCLLNNFYPREAKVQWKVDNALQSGNSQESVTEQDSKDSTYSLSSTLTLSKADYEKHKVYACEVTHQGLSSPVTKSFNRGEC
Молекула, связывающая PD-1
Последовательность гена для тяжелой цепи гибридного белка ниволумаб-галакорин (SEQ ID NO:34):
ATGATGTCCTTTGTCTCTCTGCTCCTGGTTGGCATCCTATTCCATGCCACCCAGGCC CAAGTCCAGCTCGTGGAATCAGGAGGGGGTGTAGTCCAACCAGGGCGGAGCCTCCGACTTGATTGTAAAGCATCAGGAATTACATTTTCTAATAGCGGAATGCATTGGGTCCGACAAGCGCCAGGCAAGGGACTGGAATGGGTCGCGGTGATATGGTATGATGGATCAAAACGCTATTATGCGGACTCTGTGAAGGGTCGATTCACTATTAGCAGAGATAACAGCAAGAATACTCTCTTCCTTCAGATGAATTCACTTAGGGCAGAGGACACAGCGGTGTACTATTGCGCGACGAACGACGATTATTGGGGCCAAGGGACATTGGTAACGGTGAGTTCTGCTAGTACTAAAGGGCCTTCCGTCTTCCCACTCGCCCCTTGTTCTAGAAGTACTAGTGAGTCAACAGCTGCTTTGGGTTGCTTGGTTAAAGACTACTTTCCTGAACCCGTGACTGTGTCTTGGAATTCCGGTGCTCTTACTTCAGGTGTTCATACATTCCCAGCAGTATTGCAGAGCTCTGGCTTGTATTCTCTCTCCTCAGTGGTGACAGTACCTTCCTCCTCTCTTGGCACAAAAACTTACACATGTAATGTAGACCATAAACCATCAAACACGAAAGTTGACAAGAGAGTAGAAAGCAAGTATGGGCCTCCATGTCCCCCGTGCCCGGCGCCCGAGTTCCTGGGTGGTCCGTCAGTGTTCTTGTTCCCTCCCAAGCCAAAAGATACATTGATGATAAGTCGGACGCCGGAGGTCACATGTGTAGTAGTTGATGTCTCTCAGGAGGATCCTGAGGTGCAGTTTAACTGGTACGTCGATGGTGTTGAGGTACACAACGCCAAAACTAAGCCGAGGGAAGAGCAGTTCAATTCAACATATCGGGTCGTGTCCGTATTGACAGTTCTGCACCAAGATTGGTTGAACGGAAAAGAGTATAAGTGCAAAGTTAGCAATAAGGGACTTCCGTCCTCAATTGAAAAAACCATTTCCAAAGCGAAAGGCCAACCTCGGGAACCTCAGGTATATACCTTGCCACCCAGCCAAGAAGAAATGACTAAAAACCAGGTTAGTTTGACATGTTTGGTTAAAGGCTTTTACCCGTCCGACATTGCCGTCGAGTGGGAAAGCAATGGGCAGCCTGAAAATAACTACAAGACAACCCCACCAGTATTGGATTCCGACGGTTCCTTCTTTCTTTACAGCCGCCTCACCGTCGATAAGAGTCGGTGGCAAGAGGGGAATGTCTTTTCCTGTAGTGTCATGCACGAAGCACTTCACAACCATTACACCCAAAAATCATTGTCCCTGTCACTGGGGAAA TCCGGGGGTGGCGGATCC GATGAGGCTGCAGGGATAGGCCCAGAAGTTCCTGATGACCGCGACTTCGAGCCCTCCCTAGGCCCAGTGTGCCCCTTCCGCTGTCAATGCCATCTTCGAGTGGTCCAGTGTTCTGATTTGGGTCTGGACAAAGTGCCAAAGGATCTTCCCCCTGACACAACTCTGCTAGACCTGCAAAACAACAAAATAACCGAAATCAAAGATGGAGACTTTAAGAACCTGAAGAACCTTCACGCATTGATTCTTGTCAACAATAAAATTAGCAAAGTTAGTCCTGGAGCATTTACACCTTTGGTGAAGTTGGAACGACTTTATCTGTCCAAGAATCAGCTGAAGGAATTGCCAGAAAAAATGCCCAAAACTCTTCAGGAGCTGCGTGCCCATGAGAATGAGATCACCAAAGTGCGAAAAGTTACTTTCAATGGACTGAACCAGATGATTGTCATAGAACTGGGCACCAATCCGCTGAAGAGCTCAGGAATTGAAAATGGGGCTTTCCAGGGAATGAAGAAGCTCTCCTACATCCGCATTGCTGATACCAATATCACCAGCATTCCTCAAGGTCTTCCTCCTTCCCTTACGGAATTACATCTTGATGGCAACAAAATCAGCAGAGTTGATGCAGCTAGCCTGAAAGGACTGAATAATTTGGCTAAGTTGGGATTGAGTTTCAACAGCATCTCTGCTGTTGACAATGGCTCTCTGGCCAACACGCCTCATCTGAGGGAGCTTCACTTGGACAACAACAAGCTTACCAGAGTACCTGGTGGGCTGGCAGAGCATAAGTACATCCAGGTTGTCTACCTTCATAACAACAATATCTCTGTAGTTGGATCAAGTGACTTCTGCCCACCTGGACACAACACCAAAAAGGCTTCTTATTCGGGTGTGAGTCTTTTCAGCAACCCGGTCCAGTACTGGGAGATACAGCCATCCACCTTCAGATGTGTCTACGTGCGCTCTGCCATTCAACTCGGAAACTATAAGTGA
Кодирующая сигнальный пептид последовательность выделена жирным шрифтом. Кодирующая тяжелую цепь последовательность подчеркнута прямой линией. Кодирующая линкер последовательность выделена курсивом. Кодирующая галакорин последовательность подчеркнута волнистой линией.
Последовательность белка для тяжелой цепи гибридного белка ниволумаб-галакорин (SEQ ID NO:35):
MMSFVSLLLVGILFHATQA QVQLVESGGGVVQPGRSLRLDCKASGITFSNSGMHWVRQAPGKGLEWVAVIWYDGSKRYYADSVKGRFTISRDNSKNTLFLQMNSLRAEDTAVYYCATNDDYWGQGTLVTVSSASTKGPSVFPLAPCSRSTSESTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTKTYTCNVDHKPSNTKVDKRVESKYGPPCPPCPAPEFLGGPSVFLFPPKPKDTLMISRTPEVTCVVVDVSQEDPEVQFNWYVDGVEVHNAKTKPREEQFNSTYRVVSVLTVLHQDWLNGKEYKCKVSNKGLPSSIEKTISKAKGQPREPQVYTLPPSQEEMTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSRLTVDKSRWQEGNVFSCSVMHEALHNHYTQKSLSLSLGK SGGGGS DEAAGIGPEVPDDRDFEPSLGPVCPFRCQCHLRVVQCSDLGLDKVPKDLPPDTTLLDLQNNKITEIKDGDFKNLKNLHALILVNNKISKVSPGAFTPLVKLERLYLSKNQLKELPEKMPKTLQELRAHENEITKVRKVTFNGLNQMIVIELGTNPLKSSGIENGAFQGMKKLSYIRIADTNITSIPQGLPPSLTELHLDGNKISRVDAASLKGLNNLAKLGLSFNSISAVDNGSLANTPHLRELHLDNNKLTRVPGGLAEHKYIQVVYLHNNNISVVGSSDFCPPGHNTKKASYSGVSLFSNPVQYWEIQPSTFRCVYVRSAIQLGNYK
Последовательность сигнального пептида выделена жирным шрифтом. Последовательность тяжелой цепи подчеркнута прямой линией. Последовательность линкера выделена курсивом. Последовательность галакорина подчеркнута волнистой линией.
Последовательность гена для легкой цепи ниволумаба (SEQ ID NO:36):
ATGATGTCCTTTGTCTCTCTGCTCCTGGTTGGCATCCTATTCCATGCCACCCAGGCC GAAATCGTATTGACTCAGTCCCCTGCTACACTTTCACTGAGTCCGGGTGAGCGGGCGACTTTGTCATGTCGGGCATCACAGAGTGTAAGTAGTTATCTGGCCTGGTATCAACAGAAACCGGGACAGGCTCCTCGCCTGCTGATTTATGACGCAAGCAATCGCGCGACCGGCATCCCGGCGAGGTTCTCAGGGTCTGGATCAGGTACTGACTTTACCCTTACGATCTCTTCTCTCGAACCTGAGGATTTCGCTGTCTATTACTGCCAGCAGTCTTCTAACTGGCCGAGAACATTTGGTCAAGGGACAAAAGTCGAGATTAAGCGAACTGTCGCAGCGCCATCTGTCTTTATCTTCCCTCCAAGCGACGAACAGCTTAAGAGTGGCACCGCCAGTGTTGTCTGCCTTCTGAATAACTTCTATCCAAGGGAAGCGAAAGTTCAGTGGAAGGTGGATAACGCTCTGCAGTCTGGGAACTCTCAGGAAAGTGTAACAGAACAAGACTCCAAAGACTCAACCTACTCTCTTAGTTCCACGTTGACCCTCTCCAAAGCGGACTATGAGAAGCATAAGGTCTACGCTTGCGAGGTAACACACCAGGGGCTGAGTAGTCCGGTTACGAAGAGCTTCAACAGAGGGGAATGCTGA
Кодирующая сигнальный пептид последовательность выделена жирным шрифтом. Кодирующая легкую цепь последовательность подчеркнута прямой линией.
Последовательность белка для легкой цепи ниволумаба (SEQ ID NO:37):
MMSFVSLLLVGILFHATQA EIVLTQSPATLSLSPGERATLSCRASQSVSSYLAWYQQKPGQAPRLLIYDASNRATGIPARFSGSGSGTDFTLTISSLEPEDFAVYYCQQSSNWPRTFGQGTKVEIKRTVAAPSVFIFPPSDEQLKSGTASVVCLLNNFYPREAKVQWKVDNALQSGNSQESVTEQDSKDSTYSLSSTLTLSKADYEKHKVYACEVTHQGLSSPVTKSFNRGEC
Последовательность сигнального пептида выделена жирным шрифтом. Последовательность легкой цепи подчеркнута прямой линией.
Входящие в состав аминокислотные последовательности:
Сигнальная последовательность для тяжелой и легкой цепей (SEQ ID NO:5)
Последовательность линкера между декорином и тяжелой цепью (SEQ ID NO:6)
Декорин (SEQ ID NO:7)
Последовательность белка для тяжелой цепи ниволумаба (SEQ ID NO:38):
QVQLVESGGGVVQPGRSLRLDCKASGITFSNSGMHWVRQAPGKGLEWVAVIWYDGSKRYYADSVKGRFTISRDNSKNTLFLQMNSLRAEDTAVYYCATNDDYWGQGTLVTVSSASTKGPSVFPLAPCSRSTSESTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTKTYTCNVDHKPSNTKVDKRVESKYGPPCPPCPAPEFLGGPSVFLFPPKPKDTLMISRTPEVTCVVVDVSQEDPEVQFNWYVDGVEVHNAKTKPREEQFNSTYRVVSVLTVLHQDWLNGKEYKCKVSNKGLPSSIEKTISKAKGQPREPQVYTLPPSQEEMTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSRLTVDKSRWQEGNVFSCSVMHEALHNHYTQKSLSLSLGK
Последовательность белка для легкой цепи ниволумаба (SEQ ID NO:39):
EIVLTQSPATLSLSPGERATLSCRASQSVSSYLAWYQQKPGQAPRLLIYDASNRATGIPARFSGSGSGTDFTLTISSLEPEDFAVYYCQQSSNWPRTFGQGTKVEIKRTVAAPSVFIFPPSDEQLKSGTASVVCLLNNFYPREAKVQWKVDNALQSGNSQESVTEQDSKDSTYSLSSTLTLSKADYEKHKVYACEVTHQGLSSPVTKSFNRGEC
Молекула, связывающая PD-1
Последовательность гена для тяжелой цепи гибридного белка пембролизумаб-галакорин (SEQ ID NO:40):
ATGATGTCCTTTGTCTCTCTGCTCCTGGTTGGCATCCTATTCCATGCCACCCAGGCC CAGGTGCAATTGGTCCAAAGTGGGGTCGAGGTCAAGAAGCCAGGAGCTTCTGTAAAAGTTTCATGTAAGGCATCTGGGTATACCTTCACGAACTACTATATGTATTGGGTGCGCCAAGCGCCAGGGCAGGGCCTCGAATGGATGGGTGGCATCAATCCGAGCAACGGGGGCACCAACTTTAACGAAAAGTTTAAAAACCGGGTCACCTTGACAACGGACAGTAGCACGACTACCGCTTATATGGAGCTGAAGAGTTTGCAGTTTGATGATACTGCGGTTTATTATTGTGCACGCAGAGATTATAGGTTCGACATGGGCTTCGACTACTGGGGTCAAGGTACTACGGTAACTGTATCATCTGCTAGTACAAAGGGCCCTTCCGTTTTCCCCCTCGCCCCGTGCAGCCGCTCAACGTCCGAAAGCACCGCTGCACTTGGGTGCCTTGTAAAAGACTATTTTCCAGAGCCAGTTACCGTGTCTTGGAATAGTGGCGCACTTACGTCCGGGGTGCACACTTTTCCGGCTGTCTTGCAATCCTCTGGACTCTATTCCTTGAGTAGCGTCGTAACAGTACCAAGTAGTAGTCTCGGCACCAAAACGTATACGTGCAATGTTGATCATAAGCCTAGCAACACGAAAGTTGACAAAAGAGTTGAGAGTAAATATGGACCCCCCTGTCCGCCATGCCCGGCCCCTGAATTCCTTGGGGGCCCGTCTGTATTTCTTTTCCCGCCCAAGCCGAAGGATACACTGATGATAAGCAGAACGCCTGAGGTTACCTGCGTCGTGGTCGACGTAAGCCAGGAAGATCCTGAGGTGCAATTTAATTGGTATGTGGACGGGGTCGAGGTTCATAATGCAAAAACAAAACCCCGAGAGGAGCAATTTAATTCAACGTACAGAGTCGTTAGCGTACTTACAGTGCTGCACCAGGATTGGCTCAACGGGAAGGAGTATAAGTGCAAGGTGTCTAATAAAGGTTTGCCCTCCAGTATAGAAAAAACCATCTCAAAGGCGAAAGGACAGCCTAGAGAACCTCAAGTATATACCCTCCCACCCTCCCAAGAAGAGATGACAAAGAACCAAGTGAGTCTCACATGCCTCGTCAAGGGTTTCTACCCAAGCGATATAGCCGTAGAGTGGGAATCAAATGGTCAGCCGGAGAATAACTACAAAACTACTCCGCCAGTCTTGGATAGCGACGGGTCTTTTTTCCTGTACTCTAGGCTGACGGTGGACAAGTCAAGATGGCAGGAAGGAAATGTTTTTAGCTGCTCCGTTATGCATGAGGCTCTCCACAATCATTATACACAAAAAAGTTTGTCTCTGTCATTGGGGAAA TCCGGGGGTGGCGGATCC GATGAGGCTGCAGGGATAGGCCCAGAAGTTCCTGATGACCGCGACTTCGAGCCCTCCCTAGGCCCAGTGTGCCCCTTCCGCTGTCAATGCCATCTTCGAGTGGTCCAGTGTTCTGATTTGGGTCTGGACAAAGTGCCAAAGGATCTTCCCCCTGACACAACTCTGCTAGACCTGCAAAACAACAAAATAACCGAAATCAAAGATGGAGACTTTAAGAACCTGAAGAACCTTCACGCATTGATTCTTGTCAACAATAAAATTAGCAAAGTTAGTCCTGGAGCATTTACACCTTTGGTGAAGTTGGAACGACTTTATCTGTCCAAGAATCAGCTGAAGGAATTGCCAGAAAAAATGCCCAAAACTCTTCAGGAGCTGCGTGCCCATGAGAATGAGATCACCAAAGTGCGAAAAGTTACTTTCAATGGACTGAACCAGATGATTGTCATAGAACTGGGCACCAATCCGCTGAAGAGCTCAGGAATTGAAAATGGGGCTTTCCAGGGAATGAAGAAGCTCTCCTACATCCGCATTGCTGATACCAATATCACCAGCATTCCTCAAGGTCTTCCTCCTTCCCTTACGGAATTACATCTTGATGGCAACAAAATCAGCAGAGTTGATGCAGCTAGCCTGAAAGGACTGAATAATTTGGCTAAGTTGGGATTGAGTTTCAACAGCATCTCTGCTGTTGACAATGGCTCTCTGGCCAACACGCCTCATCTGAGGGAGCTTCACTTGGACAACAACAAGCTTACCAGAGTACCTGGTGGGCTGGCAGAGCATAAGTACATCCAGGTTGTCTACCTTCATAACAACAATATCTCTGTAGTTGGATCAAGTGACTTCTGCCCACCTGGACACAACACCAAAAAGGCTTCTTATTCGGGTGTGAGTCTTTTCAGCAACCCGGTCCAGTACTGGGAGATACAGCCATCCACCTTCAGATGTGTCTACGTGCGCTCTGCCATTCAACTCGGAAACTATAAGTGA
Кодирующая сигнальный пептид последовательность выделена жирным шрифтом. Кодирующая тяжелую цепь последовательность подчеркнута прямой линией. Кодирующая линкер последовательность выделена курсивом. Кодирующая галакорин последовательность подчеркнута волнистой линией.
Последовательность белка для тяжелой цепи гибридного белка пембролизумаб-галакорин (SEQ ID NO:41):
MMSFVSLLLVGILFHATQA QVQLVQSGVEVKKPGASVKVSCKASGYTFTNYYMYWVRQAPGQGLEWMGGINPSNGGTNFNEKFKNRVTLTTDSSTTTAYMELKSLQFDDTAVYYCARRDYRFDMGFDYWGQGTTVTVSSASTKGPSVFPLAPCSRSTSESTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTKTYTCNVDHKPSNTKVDKRVESKYGPPCPPCPAPEFLGGPSVFLFPPKPKDTLMISRTPEVTCVVVDVSQEDPEVQFNWYVDGVEVHNAKTKPREEQFNSTYRVVSVLTVLHQDWLNGKEYKCKVSNKGLPSSIEKTISKAKGQPREPQVYTLPPSQEEMTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSRLTVDKSRWQEGNVFSCSVMHEALHNHYTQKSLSLSLGK SGGGGS DEAAGIGPEVPDDRDFEPSLGPVCPFRCQCHLRVVQCSDLGLDKVPKDLPPDTTLLDLQNNKITEIKDGDFKNLKNLHALILVNNKISKVSPGAFTPLVKLERLYLSKNQLKELPEKMPKTLQELRAHENEITKVRKVTFNGLNQMIVIELGTNPLKSSGIENGAFQGMKKLSYIRIADTNITSIPQGLPPSLTELHLDGNKISRVDAASLKGLNNLAKLGLSFNSISAVDNGSLANTPHLRELHLDNNKLTRVPGGLAEHKYIQVVYLHNNNISVVGSSDFCPPGHNTKKASYSGVSLFSNPVQYWEIQPSTFRCVYVRSAIQLGNYK.
Последовательность сигнального пептида выделена жирным шрифтом. Последовательность тяжелой цепи подчеркнута прямой линией. Последовательность линкера выделена курсивом. Последовательность галакорина подчеркнута волнистой линией.
Последовательность гена для легкой цепи пембролизумаба (SEQ ID NO:42):
ATGATGTCCTTTGTCTCTCTGCTCCTGGTTGGCATCCTATTCCATGCCACCCAGGCC GAAATTGTTCTTACGCAGAGTCCAGCAACTTTGTCACTCTCTCCCGGCGAACGAGCGACATTGTCCTGTCGCGCGAGTAAGGGTGTCTCAACATCTGGATACTCATATCTGCATTGGTACCAGCAAAAACCGGGACAAGCGCCGCGATTGCTGATTTATCTCGCCTCCTACCTTGAAAGTGGTGTGCCTGCGAGGTTCTCCGGTAGTGGATCAGGCACCGATTTCACTTTGACCATCAGCAGCCTCGAACCAGAAGATTTTGCCGTCTACTACTGCCAACATAGCAGGGATTTGCCACTGACATTCGGCGGGGGTACGAAAGTTGAGATTAAACGGACTGTAGCGGCACCTTCTGTCTTCATCTTCCCACCAAGCGATGAGCAGCTTAAAAGCGGTACAGCTTCAGTGGTGTGCCTTTTGAACAACTTTTATCCGCGAGAAGCCAAGGTCCAATGGAAGGTGGATAACGCTTTGCAATCCGGTAACTCACAGGAGTCAGTAACAGAGCAAGATAGTAAAGATAGCACGTATTCACTTAGCAGTACCCTTACTCTTAGCAAGGCTGATTATGAAAAACATAAGGTATATGCGTGCGAGGTAACGCACCAAGGACTTAGCTCCCCAGTGACGAAGTCATTTAACCGGGGGGAGTGCTGA
Кодирующая сигнальный пептид последовательность выделена жирным шрифтом. Кодирующая легкую цепь последовательность подчеркнута прямой линией.
Последовательность белка для легкой цепи пембролизумаба (SEQ ID NO:43):
MMSFVSLLLVGILFHATQA EIVLTQSPATLSLSPGERATLSCRASKGVSTSGYSYLHWYQQKPGQAPRLLIYLASYLESGVPARFSGSGSGTDFTLTISSLEPEDFAVYYCQHSRDLPLTFGGGTKVEIKRTVAAPSVFIFPPSDEQLKSGTASVVCLLNNFYPREAKVQWKVDNALQSGNSQESVTEQDSKDSTYSLSSTLTLSKADYEKHKVYACEVTHQGLSSPVTKSFNRGEC.
Последовательность сигнального пептида выделена жирным шрифтом. Последовательность легкой цепи подчеркнута прямой линией.
Входящие в состав аминокислотные последовательности:
Сигнальная последовательность для тяжелой и легкой цепей (SEQ ID NO:5)
Последовательность линкера между декорином и тяжелой цепью (SEQ ID NO:6)
Декорин (SEQ ID NO:7)
Последовательность белка для тяжелой цепи пембролизумаба (SEQ ID NO:44):
QVQLVQSGVEVKKPGASVKVSCKASGYTFTNYYMYWVRQAPGQGLEWMGGINPSNGGTNFNEKFKNRVTLTTDSSTTTAYMELKSLQFDDTAVYYCARRDYRFDMGFDYWGQGTTVTVSSASTKGPSVFPLAPCSRSTSESTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTKTYTCNVDHKPSNTKVDKRVESKYGPPCPPCPAPEFLGGPSVFLFPPKPKDTLMISRTPEVTCVVVDVSQEDPEVQFNWYVDGVEVHNAKTKPREEQFNSTYRVVSVLTVLHQDWLNGKEYKCKVSNKGLPSSIEKTISKAKGQPREPQVYTLPPSQEEMTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSRLTVDKSRWQEGNVFSCSVMHEALHNHYTQKSLSLSLGK
Последовательность белка для легкой цепи пембролизумаба (SEQ ID NO:45):
EIVLTQSPATLSLSPGERATLSCRASKGVSTSGYSYLHWYQQKPGQAPRLLIYLASYLESGVPARFSGSGSGTDFTLTISSLEPEDFAVYYCQHSRDLPLTFGGGTKVEIKRTVAAPSVFIFPPSDEQLKSGTASVVCLLNNFYPREAKVQWKVDNALQSGNSQESVTEQDSKDSTYSLSSTLTLSKADYEKHKVYACEVTHQGLSSPVTKSFNRGEC
Молекула, связывающая PD-L1
Последовательность гена для тяжелой цепи гибридного белка авелумаб-галакорин (SEQ ID NO:50):
AGCTTTTGGAGTCAGGCGGGGGGCTCGTCCAACCTGGGGGGTCACTCCGGTTGTCATGTGCTGCCAGTGGCTTCACATTCTCATCTTACATTATGATGTGGGTTCGACAGGCCCCTGGGAAGGGGCTGGAGTGGGTTTCCTCCATCTACCCCTCCGGGGGAATTACCTTCTATGCCGACACGGTAAAGGGTCGCTTCACTATAAGTCGAGATAACAGCAAAAATACGCTGTATCTCCAGATGAACTCTCTCAGGGCTGAGGACACAGCTGTATATTACTGCGCGCGGATTAAGTTGGGGACCGTCACAACAGTGGATTACTGGGGTCAAGGCACTCTGGTAACCGTATCCTCAGCATCCACCAAGGGCCCAAGTGTATTCCCGCTGGCCCCTTCAAGTAAATCCACGTCTGGCGGCACAGCCGCTCTCGGTTGCCTGGTTAAGGACTACTTCCCAGAACCTGTCACTGTCAGTTGGAACTCAGGCGCATTGACATCTGGTGTCCATACATTCCCCGCAGTCCTGCAAAGCTCTGGACTTTACAGTCTTAGTAGCGTAGTGACAGTCCCATCTTCAAGTCTTGGGACCCAAACTTATATTTGCAACGTAAATCATAAACCCTCCAACACTAAAGTAGACAAAAAAGTAGAGCCGAAATCTTGCGACAAAACGCATACATGCCCACCATGTCCCGCTCCGGAACTCCTGGGCGGCCCGTCCGTTTTTCTCTTTCCCCCAAAGCCCAAGGATACGCTTATGATCAGCAGAACACCGGAAGTTACTTGTGTAGTCGTTGACGTGTCTCACGAAGATCCCGAAGTCAAATTTAATTGGTATGTGGATGGCGTCGAAGTGCACAACGCAAAAACCAAACCCAGAGAGGAACAGTATAACAGCACGTATCGAGTGGTCTCCGTACTTACGGTCCTCCACCAGGACTGGTTGAATGGCAAGGAGTACAAGTGCAAAGTGAGCAATAAAGCGTTGCCAGCCCCGATCGAAAAAACCATCAGCAAGGCCAAGGGACAGCCTAGAGAGCCGCAGGTTTACACCTTGCCGCCATCAAGGGATGAACTGACTAAAAACCAGGTATCCCTGACCTGCCTGGTTAAGGGTTTTTACCCCAGTGATATAGCGGTTGAATGGGAGTCTAACGGGCAGCCAGAGAACAACTACAAAACGACACCTCCCGTTCTGGATTCCGATGGCAGCTTTTTCTTGTATTCTAAACTCACCGTGGATAAATCCCGATGGCAGCAAGGCAACGTCTTCTCCTGCAGCGTGATGCATGAAGCCTTGCACAACCACTATACCCAAAAGAGTCTCAGCCTGTCACCcGGGAAA TCCGGGGGTGGCGGATCC GATGAGGCTGCAGGGATAGGCCCAGAAGTTCCTGATGACCGCGACTTCGAGCCCTCCCTAGGCCCAGTGTGCCCCTTCCGCTGTCAATGCCATCTTCGAGTGGTCCAGTGTTCTGATTTGGGTCTGGACAAAGTGCCAAAGGATCTTCCCCCTGACACAACTCTGCTAGACCTGCAAAACAACAAAATAACCGAAATCAAAGATGGAGACTTTAAGAACCTGAAGAACCTTCACGCATTGATTCTTGTCAACAATAAAATTAGCAAAGTTAGTCCTGGAGCATTTACACCTTTGGTGAAGTTGGAACGACTTTATCTGTCCAAGAATCAGCTGAAGGAATTGCCAGAAAAAATGCCCAAAACTCTTCAGGAGCTGCGTGCCCATGAGAATGAGATCACCAAAGTGCGAAAAGTTACTTTCAATGGACTGAACCAGATGATTGTCATAGAACTGGGCACCAATCCGCTGAAGAGCTCAGGAATTGAAAATGGGGCTTTCCAGGGAATGAAGAAGCTCTCCTACATCCGCATTGCTGATACCAATATCACCAGCATTCCTCAAGGTCTTCCTCCTTCCCTTACGGAATTACATCTTGATGGCAACAAAATCAGCAGAGTTGATGCAGCTAGCCTGAAAGGACTGAATAATTTGGCTAAGTTGGGATTGAGTTTCAACAGCATCTCTGCTGTTGACAATGGCTCTCTGGCCAACACGCCTCATCTGAGGGAGCTTCACTTGGACAACAACAAGCTTACCAGAGTACCTGGTGGGCTGGCAGAGCATAAGTACATCCAGGTTGTCTACCTTCATAACAACAATATCTCTGTAGTTGGATCAAGTGACTTCTGCCCACCTGGACACAACACCAAAAAGGCTTCTTATTCGGGTGTGAGTCTTTTCAGCAACCCGGTCCAGTACTGGGAGATACAGCCATCCACCTTCAGATGTGTCTACGTGCGCTCTGCCATTCAACTCGGAAACTATAAGTGA
Кодирующая сигнальный пептид последовательность выделена жирным шрифтом. Кодирующая тяжелую цепь последовательность подчеркнута прямой линией. Кодирующая линкер последовательность выделена курсивом. Кодирующая галакорин последовательность подчеркнута волнистой линией.
Последовательность белка для тяжелой цепи гибридного белка авелумаб-галакорин (SEQ ID NO:51):
MMSFVSLLLVGILFHATQA EVQLLESGGGLVQPGGSLRLSCAASGFTFSSYIMMWVRQAPGKGLEWVSSIYPSGGITFYADTVKGRFTISRDNSKNTLYLQMNSLRAEDTAVYYCARIKLGTVTTVDYWGQGTLVTVSSASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKKVEPKSCDKTHTCPPCPAPELLGGPSVFLFPPKPKDTLMISRTPEVTCVVVDVSHEDPEVKFNWYVDGVEVHNAKTKPREEQYNSTYRVVSVLTVLHQDWLNGKEYKCKVSNKALPAPIEKTISKAKGQPREPQVYTLPPSRDELTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSKLTVDKSRWQQGNVFSCSVMHEALHNHYTQKSLSLSPGK SGGGGS DEAAGIGPEVPDDRDFEPSLGPVCPFRCQCHLRVVQCSDLGLDKVPKDLPPDTTLLDLQNNKITEIKDGDFKNLKNLHALILVNNKISKVSPGAFTPLVKLERLYLSKNQLKELPEKMPKTLQELRAHENEITKVRKVTFNGLNQMIVIELGTNPLKSSGIENGAFQGMKKLSYIRIADTNITSIPQGLPPSLTELHLDGNKISRVDAASLKGLNNLAKLGLSFNSISAVDNGSLANTPHLRELHLDNNKLTRVPGGLAEHKYIQVVYLHNNNISVVGSSDFCPPGHNTKKASYSGVSLFSNPVQYWEIQPSTFRCVYVRSAIQLGNYK.
Последовательность сигнального пептида выделена жирным шрифтом. Последовательность тяжелой цепи подчеркнута прямой линией. Последовательность линкера выделена курсивом. Последовательность галакорина подчеркнута волнистой линией.
Последовательность гена для легкой цепи авелумаба (SEQ ID NO:52):
ATGATGTCCTTTGTCTCTCTGCTCCTGGTTGGCATCCTATTCCATGCCACCCAGGCC CAGTCTGCACTTACACAACCGGCGTCCGTTTCCGGATCTCCAGGACAGAGCATTACTATCAGTTGCACGGGAACCTCCTCAGACGTAGGGGGGTATAATTATGTGTCTTGGTATCAACAGCATCCCGGGAAAGCCCCCAAACTGATGATCTACGATGTCAGCAATAGACCAAGCGGTGTGAGTAATCGATTTAGCGGGTCTAAATCTGGTAACACAGCATCCCTCACTATTAGTGGACTGCAAGCAGAAGATGAGGCAGACTATTATTGCAGTAGCTATACGTCTAGTTCCACCCGCGTTTTTGGCACTGGGACGAAAGTCACCGTTCTCGGACAACCAAAAGCAAACCCCACCGTGACTCTGTTTCCGCCTAGCAGCGAAGAATTGCAGGCCAATAAGGCGACACTCGTATGCCTTATCTCCGACTTCTACCCGGGCGCTGTGACAGTCGCGTGGAAAGCCGACGGCAGCCCTGTTAAAGCTGGAGTCGAGACCACGAAGCCGTCCAAGCAGAGTAACAATAAGTATGCTGCATCCAGTTATCTCTCTCTCACTCCGGAACAGTGGAAGTCCCATCGGTCCTATAGTTGCCAAGTGACCCATGAGGGTTCCACCGTAGAGAAAACGGTAGCACCTACCGAATGTAGTTGA
Кодирующая сигнальный пептид последовательность выделена жирным шрифтом. Кодирующая легкую цепь последовательность подчеркнута прямой линией.
Последовательность белка для легкой цепи авелумаба (SEQ ID NO:53):
MMSFVSLLLVGILFHATQA QSALTQPASVSGSPGQSITISCTGTSSDVGGYNYVSWYQQHPGKAPKLMIYDVSNRPSGVSNRFSGSKSGNTASLTISGLQAEDEADYYCSSYTSSSTRVFGTGTKVTVLGQPKANPTVTLFPPSSEELQANKATLVCLISDFYPGAVTVAWKADGSPVKAGVETTKPSKQSNNKYAASSYLSLTPEQWKSHRSYSCQVTHEGSTVEKTVAPTECS.
Последовательность сигнального пептида выделена жирным шрифтом. Последовательность легкой цепи подчеркнута прямой линией.
Молекула, связывающая PD-L1
В некоторых вариантах реализации конструкции содержат две или более копий галакорина или другой молекулы декорина, присоединенных к каждому C гена тяжелой цепи или часть молекулы галакорина (например, связывающий TGF-бета фрагмент), присоединенную к гену тяжелой цепи в виде единого целого или в виде двух или более копий. В молекуле галакорина/декорина имеется несколько связывающих TGF-бета доменов. Эти домены представлены в любой подходящей конфигурации. В некоторых вариантах реализации каждый из описанных выше вариантов присоединен к биспецифичному или полиспецифичному антителу, нацеленному на 2 или более мишени. Иллюстративные последовательности представлены ниже.
Последовательность гена для тяжелой цепи гибридного белка авелумаб-галакорин 2x (SEQ ID NO:54):
ATGATGTCCTTTGTCTCTCTGCTCCTGGTTGGCATCCTATTCCATGCCACCCAGGCC GAGGTACAGCTTTTGGAGTCAGGCGGGGGGCTCGTCCAACCTGGGGGGTCACTCCGGTTGTCATGTGCTGCCAGTGGCTTCACATTCTCATCTTACATTATGATGTGGGTTCGACAGGCCCCTGGGAAGGGGCTGGAGTGGGTTTCCTCCATCTACCCCTCCGGGGGAATTACCTTCTATGCCGACACGGTAAAGGGTCGCTTCACTATAAGTCGAGATAACAGCAAAAATACGCTGTATCTCCAGATGAACTCTCTCAGGGCTGAGGACACAGCTGTATATTACTGCGCGCGGATTAAGTTGGGGACCGTCACAACAGTGGATTACTGGGGTCAAGGCACTCTGGTAACCGTATCCTCAGCATCCACCAAGGGCCCAAGTGTATTCCCGCTGGCCCCTTCAAGTAAATCCACGTCTGGCGGCACAGCCGCTCTCGGTTGCCTGGTTAAGGACTACTTCCCAGAACCTGTCACTGTCAGTTGGAACTCAGGCGCATTGACATCTGGTGTCCATACATTCCCCGCAGTCCTGCAAAGCTCTGGACTTTACAGTCTTAGTAGCGTAGTGACAGTCCCATCTTCAAGTCTTGGGACCCAAACTTATATTTGCAACGTAAATCATAAACCCTCCAACACTAAAGTAGACAAAAAAGTAGAGCCGAAATCTTGCGACAAAACGCATACATGCCCACCATGTCCCGCTCCGGAACTCCTGGGCGGCCCGTCCGTTTTTCTCTTTCCCCCAAAGCCCAAGGATACGCTTATGATCAGCAGAACACCGGAAGTTACTTGTGTAGTCGTTGACGTGTCTCACGAAGATCCCGAAGTCAAATTTAATTGGTATGTGGATGGCGTCGAAGTGCACAACGCAAAAACCAAACCCAGAGAGGAACAGTATAACAGCACGTATCGAGTGGTCTCCGTACTTACGGTCCTCCACCAGGACTGGTTGAATGGCAAGGAGTACAAGTGCAAAGTGAGCAATAAAGCGTTGCCAGCCCCGATCGAAAAAACCATCAGCAAGGCCAAGGGACAGCCTAGAGAGCCGCAGGTTTACACCTTGCCGCCATCAAGGGATGAACTGACTAAAAACCAGGTATCCCTGACCTGCCTGGTTAAGGGTTTTTACCCCAGTGATATAGCGGTTGAATGGGAGTCTAACGGGCAGCCAGAGAACAACTACAAAACGACACCTCCCGTTCTGGATTCCGATGGCAGCTTTTTCTTGTATTCTAAACTCACCGTGGATAAATCCCGATGGCAGCAAGGCAACGTCTTCTCCTGCAGCGTGATGCATGAAGCCTTGCACAACCACTATACCCAAAAGAGTCTCAGCCTGTCACCcGGGAAA TCCGGGGGTGGCGGATCC GATGAGGCTGCAGGGATAGGCCCAGAAGTTCCTGATGACCGCGACTTCGAGCCCTCCCTAGGCCCAGTGTGCCCCTTCCGCTGTCAATGCCATCTTCGAGTGGTCCAGTGTTCTGATTTGGGTCTGGACAAAGTGCCAAAGGATCTTCCCCCTGACACAACTCTGCTAGACCTGCAAAACAACAAAATAACCGAAATCAAAGATGGAGACTTTAAGAACCTGAAGAACCTTCACGCATTGATTCTTGTCAACAATAAAATTAGCAAAGTTAGTCCTGGAGCATTTACACCTTTGGTGAAGTTGGAACGACTTTATCTGTCCAAGAATCAGCTGAAGGAATTGCCAGAAAAAATGCCCAAAACTCTTCAGGAGCTGCGTGCCCATGAGAATGAGATCACCAAAGTGCGAAAAGTTACTTTCAATGGACTGAACCAGATGATTGTCATAGAACTGGGCACCAATCCGCTGAAGAGCTCAGGAATTGAAAATGGGGCTTTCCAGGGAATGAAGAAGCTCTCCTACATCCGCATTGCTGATACCAATATCACCAGCATTCCTCAAGGTCTTCCTCCTTCCCTTACGGAATTACATCTTGATGGCAACAAAATCAGCAGAGTTGATGCAGCTAGCCTGAAAGGACTGAATAATTTGGCTAAGTTGGGATTGAGTTTCAACAGCATCTCTGCTGTTGACAATGGCTCTCTGGCCAACACGCCTCATCTGAGGGAGCTTCACTTGGACAACAACAAGCTTACCAGAGTACCTGGTGGGCTGGCAGAGCATAAGTACATCCAGGTTGTCTACCTTCATAACAACAATATCTCTGTAGTTGGATCAAGTGACTTCTGCCCACCTGGACACAACACCAAAAAGGCTTCTTATTCGGGTGTGAGTCTTTTCAGCAACCCGGTCCAGTACTGGGAGATACAGCCATCCACCTTCAGATGTGTCTACGTGCGCTCTGCCATTCAACTCGGAAACTATAAG TCCGGGGGTGGCGGATCC GATGAGGCTGCAGGGATAGGCCCAGAAGTTCCTGATGACCGCGACTTCGAGCCCTCCCTAGGCCCAGTGTGCCCCTTCCGCTGTCAATGCCATCTTCGAGTGGTCCAGTGTTCTGATTTGGGTCTGGACAAAGTGCCAAAGGATCTTCCCCCTGACACAACTCTGCTAGACCTGCAAAACAACAAAATAACCGAAATCAAAGATGGAGACTTTAAGAACCTGAAGAACCTTCACGCATTGATTCTTGTCAACAATAAAATTAGCAAAGTTAGTCCTGGAGCATTTACACCTTTGGTGAAGTTGGAACGACTTTATCTGTCCAAGAATCAGCTGAAGGAATTGCCAGAAAAAATGCCCAAAACTCTTCAGGAGCTGCGTGCCCATGAGAATGAGATCACCAAAGTGCGAAAAGTTACTTTCAATGGACTGAACCAGATGATTGTCATAGAACTGGGCACCAATCCGCTGAAGAGCTCAGGAATTGAAAATGGGGCTTTCCAGGGAATGAAGAAGCTCTCCTACATCCGCATTGCTGATACCAATATCACCAGCATTCCTCAAGGTCTTCCTCCTTCCCTTACGGAATTACATCTTGATGGCAACAAAATCAGCAGAGTTGATGCAGCTAGCCTGAAAGGACTGAATAATTTGGCTAAGTTGGGATTGAGTTTCAACAGCATCTCTGCTGTTGACAATGGCTCTCTGGCCAACACGCCTCATCTGAGGGAGCTTCACTTGGACAACAACAAGCTTACCAGAGTACCTGGTGGGCTGGCAGAGCATAAGTACATCCAGGTTGTCTACCTTCATAACAACAATATCTCTGTAGTTGGATCAAGTGACTTCTGCCCACCTGGACACAACACCAAAAAGGCTTCTTATTCGGGTGTGAGTCTTTTCAGCAACCCGGTCCAGTACTGGGAGATACAGCCATCCACCTTCAGATGTGTCTACGTGCGCTCTGCCATTCAACTCGGAAACTATAAGTGA
Кодирующая сигнальный пептид последовательность выделена жирным шрифтом. Кодирующая тяжелую цепь последовательность подчеркнута прямой линией. Кодирующие линкер последовательности выделены курсивом. Кодирующие галакорин последовательности подчеркнуты волнистой линией.
Последовательность белка для тяжелой цепи гибридного белка авелумаб-галакорин 2x (SEQ ID NO:55):
MMSFVSLLLVGILFHATQA EVQLLESGGGLVQPGGSLRLSCAASGFTFSSYIMMWVRQAPGKGLEWVSSIYPSGGITFYADTVKGRFTISRDNSKNTLYLQMNSLRAEDTAVYYCARIKLGTVTTVDYWGQGTLVTVSSASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKKVEPKSCDKTHTCPPCPAPELLGGPSVFLFPPKPKDTLMISRTPEVTCVVVDVSHEDPEVKFNWYVDGVEVHNAKTKPREEQYNSTYRVVSVLTVLHQDWLNGKEYKCKVSNKALPAPIEKTISKAKGQPREPQVYTLPPSRDELTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSKLTVDKSRWQQGNVFSCSVMHEALHNHYTQKSLSLSPGK SGGGGS DEAAGIGPEVPDDRDFEPSLGPVCPFRCQCHLRVVQCSDLGLDKVPKDLPPDTTLLDLQNNKITEIKDGDFKNLKNLHALILVNNKISKVSPGAFTPLVKLERLYLSKNQLKELPEKMPKTLQELRAHENEITKVRKVTFNGLNQMIVIELGTNPLKSSGIENGAFQGMKKLSYIRIADTNITSIPQGLPPSLTELHLDGNKISRVDAASLKGLNNLAKLGLSFNSISAVDNGSLANTPHLRELHLDNNKLTRVPGGLAEHKYIQVVYLHNNNISVVGSSDFCPPGHNTKKASYSGVSLFSNPVQYWEIQPSTFRCVYVRSAIQLGNYK SGGGGS DEAAGIGPEVPDDRDFEPSLGPVCPFRCQCHLRVVQCSDLGLDKVPKDLPPDTTLLDLQNNKITEIKDGDFKNLKNLHALILVNNKISKVSPGAFTPLVKLERLYLSKNQLKELPEKMPKTLQELRAHENEITKVRKVTFNGLNQMIVIELGTNPLKSSGIENGAFQGMKKLSYIRIADTNITSIPQGLPPSLTELHLDGNKISRVDAASLKGLNNLAKLGLSFNSISAVDNGSLANTPHLRELHLDNNKLTRVPGGLAEHKYIQVVYLHNNNISVVGSSDFCPPGHNTKKASYSGVSLFSNPVQYWEIQPSTFRCVYVRSAIQLGNYK.
Последовательность сигнального пептида выделена жирным шрифтом. Последовательность тяжелой цепи подчеркнута прямой линией. Последовательности линкеров выделены курсивом. Последовательности галакорина подчеркнуты волнистой линией.
Последовательность гена для тяжелой цепи гибридного белка авелумаб-связывающие TGF-бета домены галакорина/декорина (Asp45-Lys359 полноразмерного эндогенного декорина человека) (SEQ ID NO:56):
ATGATGTCCTTTGTCTCTCTGCTCCTGGTTGGCATCCTATTCCATGCCACCCAGGCC GAGGTACAGCTTTTGGAGTCAGGCGGGGGGCTCGTCCAACCTGGGGGGTCACTCCGGTTGTCATGTGCTGCCAGTGGCTTCACATTCTCATCTTACATTATGATGTGGGTTCGACAGGCCCCTGGGAAGGGGCTGGAGTGGGTTTCCTCCATCTACCCCTCCGGGGGAATTACCTTCTATGCCGACACGGTAAAGGGTCGCTTCACTATAAGTCGAGATAACAGCAAAAATACGCTGTATCTCCAGATGAACTCTCTCAGGGCTGAGGACACAGCTGTATATTACTGCGCGCGGATTAAGTTGGGGACCGTCACAACAGTGGATTACTGGGGTCAAGGCACTCTGGTAACCGTATCCTCAGCATCCACCAAGGGCCCAAGTGTATTCCCGCTGGCCCCTTCAAGTAAATCCACGTCTGGCGGCACAGCCGCTCTCGGTTGCCTGGTTAAGGACTACTTCCCAGAACCTGTCACTGTCAGTTGGAACTCAGGCGCATTGACATCTGGTGTCCATACATTCCCCGCAGTCCTGCAAAGCTCTGGACTTTACAGTCTTAGTAGCGTAGTGACAGTCCCATCTTCAAGTCTTGGGACCCAAACTTATATTTGCAACGTAAATCATAAACCCTCCAACACTAAAGTAGACAAAAAAGTAGAGCCGAAATCTTGCGACAAAACGCATACATGCCCACCATGTCCCGCTCCGGAACTCCTGGGCGGCCCGTCCGTTTTTCTCTTTCCCCCAAAGCCCAAGGATACGCTTATGATCAGCAGAACACCGGAAGTTACTTGTGTAGTCGTTGACGTGTCTCACGAAGATCCCGAAGTCAAATTTAATTGGTATGTGGATGGCGTCGAAGTGCACAACGCAAAAACCAAACCCAGAGAGGAACAGTATAACAGCACGTATCGAGTGGTCTCCGTACTTACGGTCCTCCACCAGGACTGGTTGAATGGCAAGGAGTACAAGTGCAAAGTGAGCAATAAAGCGTTGCCAGCCCCGATCGAAAAAACCATCAGCAAGGCCAAGGGACAGCCTAGAGAGCCGCAGGTTTACACCTTGCCGCCATCAAGGGATGAACTGACTAAAAACCAGGTATCCCTGACCTGCCTGGTTAAGGGTTTTTACCCCAGTGATATAGCGGTTGAATGGGAGTCTAACGGGCAGCCAGAGAACAACTACAAAACGACACCTCCCGTTCTGGATTCCGATGGCAGCTTTTTCTTGTATTCTAAACTCACCGTGGATAAATCCCGATGGCAGCAAGGCAACGTCTTCTCCTGCAGCGTGATGCATGAAGCCTTGCACAACCACTATACCCAAAAGAGTCTCAGCCTGTCACCcGGGAAA TCCGGGGGTGGCGGATCC GACTTCGAGCCCTCCCTAGGCCCAGTGTGCCCCTTCCGCTGTCAATGCCATCTTCGAGTGGTCCAGTGTTCTGATTTGGGTCTGGACAAAGTGCCAAAGGATCTTCCCCCTGACACAACTCTGCTAGACCTGCAAAACAACAAAATAACCGAAATCAAAGATGGAGACTTTAAGAACCTGAAGAACCTTCACGCATTGATTCTTGTCAACAATAAAATTAGCAAAGTTAGTCCTGGAGCATTTACACCTTTGGTGAAGTTGGAACGACTTTATCTGTCCAAGAATCAGCTGAAGGAATTGCCAGAAAAAATGCCCAAAACTCTTCAGGAGCTGCGTGCCCATGAGAATGAGATCACCAAAGTGCGAAAAGTTACTTTCAATGGACTGAACCAGATGATTGTCATAGAACTGGGCACCAATCCGCTGAAGAGCTCAGGAATTGAAAATGGGGCTTTCCAGGGAATGAAGAAGCTCTCCTACATCCGCATTGCTGATACCAATATCACCAGCATTCCTCAAGGTCTTCCTCCTTCCCTTACGGAATTACATCTTGATGGCAACAAAATCAGCAGAGTTGATGCAGCTAGCCTGAAAGGACTGAATAATTTGGCTAAGTTGGGATTGAGTTTCAACAGCATCTCTGCTGTTGACAATGGCTCTCTGGCCAACACGCCTCATCTGAGGGAGCTTCACTTGGACAACAACAAGCTTACCAGAGTACCTGGTGGGCTGGCAGAGCATAAGTACATCCAGGTTGTCTACCTTCATAACAACAATATCTCTGTAGTTGGATCAAGTGACTTCTGCCCACCTGGACACAACACCAAAAAGGCTTCTTATTCGGGTGTGAGTCTTTTCAGCAACCCGGTCCAGTACTGGGAGATACAGCCATCCACCTTCAGATGTGTCTACGTGCGCTCTGCCATTCAACTCGGAAACTATAAGTGA
Кодирующая сигнальный пептид последовательность выделена жирным шрифтом. Кодирующая тяжелую цепь последовательность подчеркнута прямой линией. Кодирующая линкер последовательность выделена курсивом. Кодирующая галакорин/декорин (Asp45-Lys 359) последовательность подчеркнута волнистой линией.
Последовательность белка для тяжелой цепи гибридного белка авелумаб-связывающие TGF-бета домены галакорина/декорина (Asp45-Lys359 полноразмерного эндогенного декорина человека) (SEQ ID NO:57):
MMSFVSLLLVGILFHATQA EVQLLESGGGLVQPGGSLRLSCAASGFTFSSYIMMWVRQAPGKGLEWVSSIYPSGGITFYADTVKGRFTISRDNSKNTLYLQMNSLRAEDTAVYYCARIKLGTVTTVDYWGQGTLVTVSSASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKKVEPKSCDKTHTCPPCPAPELLGGPSVFLFPPKPKDTLMISRTPEVTCVVVDVSHEDPEVKFNWYVDGVEVHNAKTKPREEQYNSTYRVVSVLTVLHQDWLNGKEYKCKVSNKALPAPIEKTISKAKGQPREPQVYTLPPSRDELTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSKLTVDKSRWQQGNVFSCSVMHEALHNHYTQKSLSLSPGK SGGGGS DFEPSLGPVCPFRCQCHLRVVQCSDLGLDKVPKDLPPDTTLLDLQNNKITEIKDGDFKNLKNLHALILVNNKISKVSPGAFTPLVKLERLYLSKNQLKELPEKMPKTLQELRAHENEITKVRKVTFNGLNQMIVIELGTNPLKSSGIENGAFQGMKKLSYIRIADTNITSIPQGLPPSLTELHLDGNKISRVDAASLKGLNNLAKLGLSFNSISAVDNGSLANTPHLRELHLDNNKLTRVPGGLAEHKYIQVVYLHNNNISVVGSSDFCPPGHNTKKASYSGVSLFSNPVQYWEIQPSTFRCVYVRSAIQLGNYK.
Последовательность сигнального пептида выделена жирным шрифтом. Последовательность тяжелой цепи подчеркнута прямой линией. Последовательность линкера выделена курсивом. Последовательность галакорина/декорина (Asp45-Lys 359) подчеркнута волнистой линией.
Последовательность гена для тяжелой цепи гибридного белка авелумаб-связывающие TGF-бета домены галакорина/декорина (Asp45-Lys359 полноразмерного эндогенного декорина человека) 2x (SEQ ID NO:58):
ATGATGTCCTTTGTCTCTCTGCTCCTGGTTGGCATCCTATTCCATGCCACCCAGGCC GAGGTACAGCTTTTGGAGTCAGGCGGGGGGCTCGTCCAACCTGGGGGGTCACTCCGGTTGTCATGTGCTGCCAGTGGCTTCACATTCTCATCTTACATTATGATGTGGGTTCGACAGGCCCCTGGGAAGGGGCTGGAGTGGGTTTCCTCCATCTACCCCTCCGGGGGAATTACCTTCTATGCCGACACGGTAAAGGGTCGCTTCACTATAAGTCGAGATAACAGCAAAAATACGCTGTATCTCCAGATGAACTCTCTCAGGGCTGAGGACACAGCTGTATATTACTGCGCGCGGATTAAGTTGGGGACCGTCACAACAGTGGATTACTGGGGTCAAGGCACTCTGGTAACCGTATCCTCAGCATCCACCAAGGGCCCAAGTGTATTCCCGCTGGCCCCTTCAAGTAAATCCACGTCTGGCGGCACAGCCGCTCTCGGTTGCCTGGTTAAGGACTACTTCCCAGAACCTGTCACTGTCAGTTGGAACTCAGGCGCATTGACATCTGGTGTCCATACATTCCCCGCAGTCCTGCAAAGCTCTGGACTTTACAGTCTTAGTAGCGTAGTGACAGTCCCATCTTCAAGTCTTGGGACCCAAACTTATATTTGCAACGTAAATCATAAACCCTCCAACACTAAAGTAGACAAAAAAGTAGAGCCGAAATCTTGCGACAAAACGCATACATGCCCACCATGTCCCGCTCCGGAACTCCTGGGCGGCCCGTCCGTTTTTCTCTTTCCCCCAAAGCCCAAGGATACGCTTATGATCAGCAGAACACCGGAAGTTACTTGTGTAGTCGTTGACGTGTCTCACGAAGATCCCGAAGTCAAATTTAATTGGTATGTGGATGGCGTCGAAGTGCACAACGCAAAAACCAAACCCAGAGAGGAACAGTATAACAGCACGTATCGAGTGGTCTCCGTACTTACGGTCCTCCACCAGGACTGGTTGAATGGCAAGGAGTACAAGTGCAAAGTGAGCAATAAAGCGTTGCCAGCCCCGATCGAAAAAACCATCAGCAAGGCCAAGGGACAGCCTAGAGAGCCGCAGGTTTACACCTTGCCGCCATCAAGGGATGAACTGACTAAAAACCAGGTATCCCTGACCTGCCTGGTTAAGGGTTTTTACCCCAGTGATATAGCGGTTGAATGGGAGTCTAACGGGCAGCCAGAGAACAACTACAAAACGACACCTCCCGTTCTGGATTCCGATGGCAGCTTTTTCTTGTATTCTAAACTCACCGTGGATAAATCCCGATGGCAGCAAGGCAACGTCTTCTCCTGCAGCGTGATGCATGAAGCCTTGCACAACCACTATACCCAAAAGAGTCTCAGCCTGTCACCcGGGAAA TCCGGGGGTGGCGGATCC GACTTCGAGCCCTCCCTAGGCCCAGTGTGCCCCTTCCGCTGTCAATGCCATCTTCGAGTGGTCCAGTGTTCTGATTTGGGTCTGGACAAAGTGCCAAAGGATCTTCCCCCTGACACAACTCTGCTAGACCTGCAAAACAACAAAATAACCGAAATCAAAGATGGAGACTTTAAGAACCTGAAGAACCTTCACGCATTGATTCTTGTCAACAATAAAATTAGCAAAGTTAGTCCTGGAGCATTTACACCTTTGGTGAAGTTGGAACGACTTTATCTGTCCAAGAATCAGCTGAAGGAATTGCCAGAAAAAATGCCCAAAACTCTTCAGGAGCTGCGTGCCCATGAGAATGAGATCACCAAAGTGCGAAAAGTTACTTTCAATGGACTGAACCAGATGATTGTCATAGAACTGGGCACCAATCCGCTGAAGAGCTCAGGAATTGAAAATGGGGCTTTCCAGGGAATGAAGAAGCTCTCCTACATCCGCATTGCTGATACCAATATCACCAGCATTCCTCAAGGTCTTCCTCCTTCCCTTACGGAATTACATCTTGATGGCAACAAAATCAGCAGAGTTGATGCAGCTAGCCTGAAAGGACTGAATAATTTGGCTAAGTTGGGATTGAGTTTCAACAGCATCTCTGCTGTTGACAATGGCTCTCTGGCCAACACGCCTCATCTGAGGGAGCTTCACTTGGACAACAACAAGCTTACCAGAGTACCTGGTGGGCTGGCAGAGCATAAGTACATCCAGGTTGTCTACCTTCATAACAACAATATCTCTGTAGTTGGATCAAGTGACTTCTGCCCACCTGGACACAACACCAAAAAGGCTTCTTATTCGGGTGTGAGTCTTTTCAGCAACCCGGTCCAGTACTGGGAGATACAGCCATCCACCTTCAGATGTGTCTACGTGCGCTCTGCCATTCAACTCGGAAACTATAAG TCCGGGGGTGGCGGATCC GACTTCGAGCCCTCCCTAGGCCCAGTGTGCCCCTTCCGCTGTCAATGCCATCTTCGAGTGGTCCAGTGTTCTGATTTGGGTCTGGACAAAGTGCCAAAGGATCTTCCCCCTGACACAACTCTGCTAGACCTGCAAAACAACAAAATAACCGAAATCAAAGATGGAGACTTTAAGAACCTGAAGAACCTTCACGCATTGATTCTTGTCAACAATAAAATTAGCAAAGTTAGTCCTGGAGCATTTACACCTTTGGTGAAGTTGGAACGACTTTATCTGTCCAAGAATCAGCTGAAGGAATTGCCAGAAAAAATGCCCAAAACTCTTCAGGAGCTGCGTGCCCATGAGAATGAGATCACCAAAGTGCGAAAAGTTACTTTCAATGGACTGAACCAGATGATTGTCATAGAACTGGGCACCAATCCGCTGAAGAGCTCAGGAATTGAAAATGGGGCTTTCCAGGGAATGAAGAAGCTCTCCTACATCCGCATTGCTGATACCAATATCACCAGCATTCCTCAAGGTCTTCCTCCTTCCCTTACGGAATTACATCTTGATGGCAACAAAATCAGCAGAGTTGATGCAGCTAGCCTGAAAGGACTGAATAATTTGGCTAAGTTGGGATTGAGTTTCAACAGCATCTCTGCTGTTGACAATGGCTCTCTGGCCAACACGCCTCATCTGAGGGAGCTTCACTTGGACAACAACAAGCTTACCAGAGTACCTGGTGGGCTGGCAGAGCATAAGTACATCCAGGTTGTCTACCTTCATAACAACAATATCTCTGTAGTTGGATCAAGTGACTTCTGCCCACCTGGACACAACACCAAAAAGGCTTCTTATTCGGGTGTGAGTCTTTTCAGCAACCCGGTCCAGTACTGGGAGATACAGCCATCCACCTTCAGATGTGTCTACGTGCGCTCTGCCATTCAACTCGGAAACTATAAGTGA
Кодирующая сигнальный пептид последовательность выделена жирным шрифтом. Кодирующая тяжелую цепь последовательность подчеркнута прямой линией. Кодирующие линкер последовательности выделены курсивом. Кодирующие галакорин/декорин (Asp45-Lys 359) последовательности подчеркнуты волнистой линией.
Последовательность белка для тяжелой цепи гибридного белка авелумаб-связывающие TGF-бета домены галакорина/декорина (Asp45-Lys359 полноразмерного эндогенного декорина человека) 2x (SEQ ID NO:59):
MMSFVSLLLVGILFHATQA EVQLLESGGGLVQPGGSLRLSCAASGFTFSSYIMMWVRQAPGKGLEWVSSIYPSGGITFYADTVKGRFTISRDNSKNTLYLQMNSLRAEDTAVYYCARIKLGTVTTVDYWGQGTLVTVSSASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKKVEPKSCDKTHTCPPCPAPELLGGPSVFLFPPKPKDTLMISRTPEVTCVVVDVSHEDPEVKFNWYVDGVEVHNAKTKPREEQYNSTYRVVSVLTVLHQDWLNGKEYKCKVSNKALPAPIEKTISKAKGQPREPQVYTLPPSRDELTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSKLTVDKSRWQQGNVFSCSVMHEALHNHYTQKSLSLSPGK SGGGGS DFEPSLGPVCPFRCQCHLRVVQCSDLGLDKVPKDLPPDTTLLDLQNNKITEIKDGDFKNLKNLHALILVNNKISKVSPGAFTPLVKLERLYLSKNQLKELPEKMPKTLQELRAHENEITKVRKVTFNGLNQMIVIELGTNPLKSSGIENGAFQGMKKLSYIRIADTNITSIPQGLPPSLTELHLDGNKISRVDAASLKGLNNLAKLGLSFNSISAVDNGSLANTPHLRELHLDNNKLTRVPGGLAEHKYIQVVYLHNNNISVVGSSDFCPPGHNTKKASYSGVSLFSNPVQYWEIQPSTFRCVYVRSAIQLGNYK SGGGGS DFEPSLGPVCPFRCQCHLRVVQCSDLGLDKVPKDLPPDTTLLDLQNNKITEIKDGDFKNLKNLHALILVNNKISKVSPGAFTPLVKLERLYLSKNQLKELPEKMPKTLQELRAHENEITKVRKVTFNGLNQMIVIELGTNPLKSSGIENGAFQGMKKLSYIRIADTNITSIPQGLPPSLTELHLDGNKISRVDAASLKGLNNLAKLGLSFNSISAVDNGSLANTPHLRELHLDNNKLTRVPGGLAEHKYIQVVYLHNNNISVVGSSDFCPPGHNTKKASYSGVSLFSNPVQYWEIQPSTFRCVYVRSAIQLGNYK.
Последовательность сигнального пептида выделена жирным шрифтом. Последовательность тяжелой цепи подчеркнута прямой линией. Последовательности линкеров выделены курсивом. Последовательности галакорина/декорина (Asp45-Lys 359) подчеркнуты волнистой линией.
Последовательность гена для тяжелой цепи гибридного белка авелумаб-связывающий TGF-бета домен галакорина/декорина (Leu155-Val260 полноразмерного эндогенного декорина человека) (SEQ ID NO:60):
ATGATGTCCTTTGTCTCTCTGCTCCTGGTTGGCATCCTATTCCATGCCACCCAGGCC GAGGTACAGCTTTTGGAGTCAGGCGGGGGGCTCGTCCAACCTGGGGGGTCACTCCGGTTGTCATGTGCTGCCAGTGGCTTCACATTCTCATCTTACATTATGATGTGGGTTCGACAGGCCCCTGGGAAGGGGCTGGAGTGGGTTTCCTCCATCTACCCCTCCGGGGGAATTACCTTCTATGCCGACACGGTAAAGGGTCGCTTCACTATAAGTCGAGATAACAGCAAAAATACGCTGTATCTCCAGATGAACTCTCTCAGGGCTGAGGACACAGCTGTATATTACTGCGCGCGGATTAAGTTGGGGACCGTCACAACAGTGGATTACTGGGGTCAAGGCACTCTGGTAACCGTATCCTCAGCATCCACCAAGGGCCCAAGTGTATTCCCGCTGGCCCCTTCAAGTAAATCCACGTCTGGCGGCACAGCCGCTCTCGGTTGCCTGGTTAAGGACTACTTCCCAGAACCTGTCACTGTCAGTTGGAACTCAGGCGCATTGACATCTGGTGTCCATACATTCCCCGCAGTCCTGCAAAGCTCTGGACTTTACAGTCTTAGTAGCGTAGTGACAGTCCCATCTTCAAGTCTTGGGACCCAAACTTATATTTGCAACGTAAATCATAAACCCTCCAACACTAAAGTAGACAAAAAAGTAGAGCCGAAATCTTGCGACAAAACGCATACATGCCCACCATGTCCCGCTCCGGAACTCCTGGGCGGCCCGTCCGTTTTTCTCTTTCCCCCAAAGCCCAAGGATACGCTTATGATCAGCAGAACACCGGAAGTTACTTGTGTAGTCGTTGACGTGTCTCACGAAGATCCCGAAGTCAAATTTAATTGGTATGTGGATGGCGTCGAAGTGCACAACGCAAAAACCAAACCCAGAGAGGAACAGTATAACAGCACGTATCGAGTGGTCTCCGTACTTACGGTCCTCCACCAGGACTGGTTGAATGGCAAGGAGTACAAGTGCAAAGTGAGCAATAAAGCGTTGCCAGCCCCGATCGAAAAAACCATCAGCAAGGCCAAGGGACAGCCTAGAGAGCCGCAGGTTTACACCTTGCCGCCATCAAGGGATGAACTGACTAAAAACCAGGTATCCCTGACCTGCCTGGTTAAGGGTTTTTACCCCAGTGATATAGCGGTTGAATGGGAGTCTAACGGGCAGCCAGAGAACAACTACAAAACGACACCTCCCGTTCTGGATTCCGATGGCAGCTTTTTCTTGTATTCTAAACTCACCGTGGATAAATCCCGATGGCAGCAAGGCAACGTCTTCTCCTGCAGCGTGATGCATGAAGCCTTGCACAACCACTATACCCAAAAGAGTCTCAGCCTGTCACCcGGGAAA TCCGGGGGTGGCGGATCC CTGCGTGCCCATGAGAATGAGATCACCAAAGTGCGAAAAGTTACTTTCAATGGACTGAACCAGATGATTGTCATAGAACTGGGCACCAATCCGCTGAAGAGCTCAGGAATTGAAAATGGGGCTTTCCAGGGAATGAAGAAGCTCTCCTACATCCGCATTGCTGATACCAATATCACCAGCATTCCTCAAGGTCTTCCTCCTTCCCTTACGGAATTACATCTTGATGGCAACAAAATCAGCAGAGTTGATGCAGCTAGCCTGAAAGGACTGAATAATTTGGCTAAGTTGGGATTGAGTTTCAACAGCATCTCTGCTGTTTGA
Кодирующая сигнальный пептид последовательность выделена жирным шрифтом. Кодирующая тяжелую цепь последовательность подчеркнута прямой линией. Кодирующая линкер последовательность выделена курсивом. Кодирующая галакорин/декорин (Leu155-Val260) последовательность подчеркнута волнистой линией.
Последовательность белка для тяжелой цепи гибридного белка авелумаб-связывающий TGF-бета домен галакорина/декорина (Leu155-Val260 полноразмерного эндогенного декорина человека) (SEQ ID NO:61):
MMSFVSLLLVGILFHATQA EVQLLESGGGLVQPGGSLRLSCAASGFTFSSYIMMWVRQAPGKGLEWVSSIYPSGGITFYADTVKGRFTISRDNSKNTLYLQMNSLRAEDTAVYYCARIKLGTVTTVDYWGQGTLVTVSSASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKKVEPKSCDKTHTCPPCPAPELLGGPSVFLFPPKPKDTLMISRTPEVTCVVVDVSHEDPEVKFNWYVDGVEVHNAKTKPREEQYNSTYRVVSVLTVLHQDWLNGKEYKCKVSNKALPAPIEKTISKAKGQPREPQVYTLPPSRDELTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSKLTVDKSRWQQGNVFSCSVMHEALHNHYTQKSLSLSPGK SGGGGS LRAHENEITKVRKVTFNGLNQMIVIELGTNPLKSSGIENGAFQGMKKLSYIRIADTNITSIPQGLPPSLTELHLDGNKISRVDAASLKGLNNLAKLGLSFNSISAV.
Последовательность сигнального пептида выделена жирным шрифтом. Последовательность тяжелой цепи подчеркнута прямой линией. Последовательность линкера выделена курсивом. Последовательность галакорина/декорина (Leu155-Val260) подчеркнута волнистой линией.
Последовательность гена для тяжелой цепи гибридного белка авелумаб-связывающий TGF-бета домен галакорина/декорина (Leu155-Val260 полноразмерного эндогенного декорина человека) 2x (SEQ ID NO:62):
ATGATGTCCTTTGTCTCTCTGCTCCTGGTTGGCATCCTATTCCATGCCACCCAGGCC GAGGTACAGCTTTTGGAGTCAGGCGGGGGGCTCGTCCAACCTGGGGGGTCACTCCGGTTGTCATGTGCTGCCAGTGGCTTCACATTCTCATCTTACATTATGATGTGGGTTCGACAGGCCCCTGGGAAGGGGCTGGAGTGGGTTTCCTCCATCTACCCCTCCGGGGGAATTACCTTCTATGCCGACACGGTAAAGGGTCGCTTCACTATAAGTCGAGATAACAGCAAAAATACGCTGTATCTCCAGATGAACTCTCTCAGGGCTGAGGACACAGCTGTATATTACTGCGCGCGGATTAAGTTGGGGACCGTCACAACAGTGGATTACTGGGGTCAAGGCACTCTGGTAACCGTATCCTCAGCATCCACCAAGGGCCCAAGTGTATTCCCGCTGGCCCCTTCAAGTAAATCCACGTCTGGCGGCACAGCCGCTCTCGGTTGCCTGGTTAAGGACTACTTCCCAGAACCTGTCACTGTCAGTTGGAACTCAGGCGCATTGACATCTGGTGTCCATACATTCCCCGCAGTCCTGCAAAGCTCTGGACTTTACAGTCTTAGTAGCGTAGTGACAGTCCCATCTTCAAGTCTTGGGACCCAAACTTATATTTGCAACGTAAATCATAAACCCTCCAACACTAAAGTAGACAAAAAAGTAGAGCCGAAATCTTGCGACAAAACGCATACATGCCCACCATGTCCCGCTCCGGAACTCCTGGGCGGCCCGTCCGTTTTTCTCTTTCCCCCAAAGCCCAAGGATACGCTTATGATCAGCAGAACACCGGAAGTTACTTGTGTAGTCGTTGACGTGTCTCACGAAGATCCCGAAGTCAAATTTAATTGGTATGTGGATGGCGTCGAAGTGCACAACGCAAAAACCAAACCCAGAGAGGAACAGTATAACAGCACGTATCGAGTGGTCTCCGTACTTACGGTCCTCCACCAGGACTGGTTGAATGGCAAGGAGTACAAGTGCAAAGTGAGCAATAAAGCGTTGCCAGCCCCGATCGAAAAAACCATCAGCAAGGCCAAGGGACAGCCTAGAGAGCCGCAGGTTTACACCTTGCCGCCATCAAGGGATGAACTGACTAAAAACCAGGTATCCCTGACCTGCCTGGTTAAGGGTTTTTACCCCAGTGATATAGCGGTTGAATGGGAGTCTAACGGGCAGCCAGAGAACAACTACAAAACGACACCTCCCGTTCTGGATTCCGATGGCAGCTTTTTCTTGTATTCTAAACTCACCGTGGATAAATCCCGATGGCAGCAAGGCAACGTCTTCTCCTGCAGCGTGATGCATGAAGCCTTGCACAACCACTATACCCAAAAGAGTCTCAGCCTGTCACCcGGGAAA TCCGGGGGTGGCGGATCC CTGCGTGCCCATGAGAATGAGATCACCAAAGTGCGAAAAGTTACTTTCAATGGACTGAACCAGATGATTGTCATAGAACTGGGCACCAATCCGCTGAAGAGCTCAGGAATTGAAAATGGGGCTTTCCAGGGAATGAAGAAGCTCTCCTACATCCGCATTGCTGATACCAATATCACCAGCATTCCTCAAGGTCTTCCTCCTTCCCTTACGGAATTACATCTTGATGGCAACAAAATCAGCAGAGTTGATGCAGCTAGCCTGAAAGGACTGAATAATTTGGCTAAGTTGGGATTGAGTTTCAACAGCATCTCTGCTGTT TCCGGGGGTGGCGGATCC CTGCGTGCCCATGAGAATGAGATCACCAAAGTGCGAAAAGTTACTTTCAATGGACTGAACCAGATGATTGTCATAGAACTGGGCACCAATCCGCTGAAGAGCTCAGGAATTGAAAATGGGGCTTTCCAGGGAATGAAGAAGCTCTCCTACATCCGCATTGCTGATACCAATATCACCAGCATTCCTCAAGGTCTTCCTCCTTCCCTTACGGAATTACATCTTGATGGCAACAAAATCAGCAGAGTTGATGCAGCTAGCCTGAAAGGACTGAATAATTTGGCTAAGTTGGGATTGAGTTTCAACAGCATCTCTGCTGTTTGA
Кодирующая сигнальный пептид последовательность выделена жирным шрифтом. Кодирующая тяжелую цепь последовательность подчеркнута прямой линией. Кодирующие линкер последовательности выделены курсивом. Кодирующие галакорин/декорин (Leu155-Val260) последовательности подчеркнуты волнистой линией.
Последовательность белка для тяжелой цепи гибридного белка авелумаб-связывающий TGF-бета домен галакорина/декорина (Leu155-Val260 полноразмерного эндогенного декорина человека) 2x (SEQ ID NO:63):
MMSFVSLLLVGILFHATQA EVQLLESGGGLVQPGGSLRLSCAASGFTFSSYIMMWVRQAPGKGLEWVSSIYPSGGITFYADTVKGRFTISRDNSKNTLYLQMNSLRAEDTAVYYCARIKLGTVTTVDYWGQGTLVTVSSASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKKVEPKSCDKTHTCPPCPAPELLGGPSVFLFPPKPKDTLMISRTPEVTCVVVDVSHEDPEVKFNWYVDGVEVHNAKTKPREEQYNSTYRVVSVLTVLHQDWLNGKEYKCKVSNKALPAPIEKTISKAKGQPREPQVYTLPPSRDELTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSKLTVDKSRWQQGNVFSCSVMHEALHNHYTQKSLSLSPGK SGGGGS LRAHENEITKVRKVTFNGLNQMIVIELGTNPLKSSGIENGAFQGMKKLSYIRIADTNITSIPQGLPPSLTELHLDGNKISRVDAASLKGLNNLAKLGLSFNSISAV SGGGGS LRAHENEITKVRKVTFNGLNQMIVIELGTNPLKSSGIENGAFQGMKKLSYIRIADTNITSIPQGLPPSLTELHLDGNKISRVDAASLKGLNNLAKLGLSFNSISAV.
Последовательность сигнального пептида выделена жирным шрифтом. Последовательность тяжелой цепи подчеркнута прямой линией. Последовательности линкеров выделены курсивом. Последовательности галакорина/декорина (Leu155-Val260) подчеркнуты волнистой линией.
Пример 2
В этом примере описано получение экспрессионных линий клеток для получения гибридных белков, описанных в примере 1.
Получение ретровектора: приведенные выше экспрессионные конструкции вводили в линию клеток HEK 293, которая конститутивно продуцирует белки gag, pro и pol MLV (Moloney murine leukemia virus, вирус лейкоза мышей Молони). Содержащую оболочку экспрессионную плазмиду также трансфицировали совместно с генетической конструкцией либо легкой цепи бевацизумаба (см. схему вектора на фигуре 2), либо гибридного белка тяжелая цепь бевацизумаба-галакорин (см. схему вектора на фигуре 3). Две указанные совместные трансфекции приводили к получению неспособного к репликации ретровектора с высоким титром либо для легкой цепи, либо для тяжелой цепи-галакорина, который концентрировали путем ультрацентрифугирования и применяли для трансдукции клеток (см., например, Bleck, G.T. 2005 An alternative method for the rapid generation of stable, high-expressing mammalian cell lines (A Technical Review). Bioprocessing J. Setp/Oct. pp 1-7; Bleck, G.T., 2010. GPEx® A Flexible Method for the Rapid Generation of Stable, High Expressing, Antibody Producing Mammalian Cell Lines Chapter 4 In: Current Trends in Monoclonal Antibody Development and Manufacturing, Biotechnology: Pharmaceutical Aspects, Edited by: S.J. Shire et al. © 2010 American Association of Pharmaceutical Scientists, DOI 10.1007/978-0-387-76643-0_4).
Сводная таблица. Характеристики pCS-Bi-wayLC-WPRE(new ori), GDD2107.0003
142 - 766 п.о. промотор hCMV
767 - 911 п.о. R-U5 MoMuSV
Промотор hCMV теряется после стадии упаковки в клетки.
981 - 1790 п.о.
1822 - 2424 п.о.
2510 - 3211 п.о.
3230 - 3830 п.о.
3870 - 4463 п.о.
5765 < 6625 п.о.
1 - 148 п.о.
Эти области теряются после трансфекции плазмид в упаковывающие клетки и получения частиц ретровектора.
Сводная таблица: характеристики pFCS-BiwayGalacorin-WPRE-SIN (new ori), GDD2134.0001
(R-U5)
149 - 865 п.о. промотор hCMV
866 - 1041 п.о. R-U5 MoMuSV
Промотор hCMV теряется после стадии упаковки в клетки.
1111 - 1920 п.о.
1952 - 2624 п.о.
2650 - 5073 п.о.
5089 - 5689 п.о.
5730 - 6153 п.о.
7472 - 8332 п.о.
1 - 148 п.о.
Эти области теряются после трансфекции плазмид в упаковывающие клетки и получения частиц ретровектора.
Трансдукция клеток GCHO с применением ретровектора : Объединенную линию клеток, продуцирующих гибридное антитело бевацизумаб-галакорин, получали с помощью нескольких циклов клеточной трансдукции родительской линии клеток яичника китайского хомячка (GPEx® Chinese Hamster Ovary, GCHO), включающих две трансдукции для легкой цепи с последующими двумя трансдукциями для молекулы тяжелая цепь-галакорин, выполняемыми один раз в неделю на протяжении 4 недель. Эти трансдукции выполняли для получения объединенной линии клеток каждого из двух генных продуктов.
Получение гибридного белка бевацизумаб-галакорин из объединенной популяции клеток, выполняемое с периодическим добавлением субстрата : после трансдукции осуществляли масштабирование для повышения продуктивности объединенной линии клеток, продуцирующих гибридный белок бевацизумаб-галакорин, в исследовании с периодическим добавлением субстрата во встряхиваемых колбах объемом 250 мл в дупликатах. В каждую встряхиваемую колбу высевали 300000 жизнеспособных клеток на мл в рабочем объеме 60 мл среды PF CHO LS (HyClone) и инкубировали в увлажненном (70-80%) инкубаторе со встряхиванием при 130 об./мин при 5% CO2 и температуре 37°C. Культуры подпитывали четыре раза в течение технологического цикла с применением двух различных питательных добавок. Культивирование прекращали, когда жизнеспособность составляла ≤ 70%. Подтверждение продукции гибридного антитела выполняли с помощью анализа методом электрофореза в полиакриламидном геле с добавлением ДСН (SDS-PAGE) (фигура 4) и с помощью ELISA для количественной оценки количества полученного продукта. Культуры продуцируют 360 мг/л продукта гибридного антитела. Поведение продукта в SDS-PAGE соответствовало ожидаемому, наблюдали преобладающую одиночную зону в невосстанавливающих условиях и две зоны (гибридного белка тяжелая цепь-галакорин и легкой цепи) в восстанавливающих условиях. Размер гибридного белка тяжелой цепи ~ 80 кДа соответствует ожидаемому размеру этого продукта, и легкая цепь имеет нормальный размер `25 кДа.
Пример 3
В этом примере описана экспрессия гибридного белка авелумаб-галакорин.
Генетическую конструкцию SEQ ID NO:50 временно трансфицировали в клетки ExpiCHO, и получение гибридной молекулы проводили в масштабе 250 мл. Титр гибридного белка в день сбора составлял 377 мг/л. Полученный гибридный белок очищали на колонке с белком A MabSelectSuRe. Полученный очищенный белок переводили в буфер, содержащий 20 мМ цитрат натрия, pH 5,5, 50 мМ NaCl, с помощью тангенциальной поточной фильтрации. Полученный продукт исследовали с помощью электрофореза в гелях SDS-PAGE и эксклюзионной ВЭЖХ. Продукт демонстрировал ожидаемые характеристики размеров в геле SDS-PAGE (ФИГ. 5) и очень низкие уровни агрегации при эксклюзионной ВЭЖХ (ФИГ. 6).
Полученный приведенным выше способом продукт применяли для оценки в исследовании на модели опухоли у мышей. В указанном исследовании использовали мышей C57BL/6 и клетки колоректального рака человека MC-38. Сорока мышам C57BL/6 вводили клетки MC-38 путем подкожной инъекции. Опухолям позволяли вырасти до примерно 100 мм3 и затем каждым 10 мышам назначали 4 различных вида лечения. Каждая группа получала однократную дозу препарата в/в, и в последующие дни определяли размер опухоли. Группы лечения включали носитель, галакорин/декорин в дозе 4 мг/кг, авелумаб/связывающую PD-L1 молекулу в дозе 17 мг/кг или гибридный белок авелумаб/связывающая PD-L1 молекула - декорин/галакорин в дозе 25 мг/кг.
Рост опухоли подавлялся введением как авелумаба/связывающей PD-L1 молекулы, так и гибридного белка авелумаб/связывающая PD-L1 молекула - декорин/галакорин по сравнению с носителем и декорином/галакорином отдельно (ФИГ. 7). Помимо этого, введение гибридного белка авелумаб/связывающая PD-L1 молекула - декорин/галакорин также подавляло рост более эффективно, чем авелумаб/связывающая PD-L1 молекула отдельно.
Все публикации и патенты, упомянутые в приведенном выше описании, включены в настоящий документ посредством ссылки. Специалистам в данной области техники будут очевидны различные модификации и варианты описанных способа и системы согласно настоящему изобретению без отступления от объема и сущности настоящего изобретения. Несмотря на то, что настоящее изобретение было описано в связи с конкретными предпочтительными вариантами реализации, следует понимать, что заявленное изобретение не должно быть неоправданно ограничено такими конкретными вариантами реализации. Действительно, предполагается, что различные модификации описанных способов практического осуществления настоящего изобретения, очевидные для специалистов в области техники, к которой относится настоящее изобретение, находятся в пределах объема приведенной ниже формулы изобретения.
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> CATALENT PHARMA SOLUTIONS, LLC
<120> MULTIFUNCTIONAL PROTEIN MOLECULES COMPRISING DECORIN
AND USE THEREOF
<130> GALA-33890
<150> US 62/693,766
<151> 2018-07-03
<160> 66
<170> PatentIn version 3.5
<210> 1
<211> 2424
<212> DNA
<213> Artificial Sequence
<220>
<223> synthetic
<400> 1
atgatgtcct ttgtctctct gctcctggtt ggcatcctat tccatgccac ccaggccgag 60
gtgcagctgg tggagtctgg gggaggcttg gtacagcctg gggggtccct gagactctcc 120
tgtgcagcct ctggatacac ctttaccaac tatggcatga actgggtccg ccaggctcca 180
gggaaggggc tggagtgggt gggctggata aacacttaca ctggtgagcc aacatatgca 240
gctgacttca agcgccggtt taccttctct ttggacacct ccaagtccac ggcctatctg 300
caaatgaaca gcctgcgggc cgaggacacg gccgtatatt actgtgcgaa atacccccac 360
tactacggta gtagccactg gtactttgac gtgtggggcc agggaaccct ggtcaccgtc 420
tcctcagcct ccaccaaggg cccatcggtc ttccccctgg caccctcctc caagagcacc 480
tctgggggca cagcggccct gggctgcctg gtcaaggact acttccccga accggtgacg 540
gtgtcgtgga actcaggcgc cctgaccagc ggcgtgcaca ccttcccggc tgtcctacag 600
tcctcaggac tctactccct cagcagcgtg gtgaccgtgc cctccagcag cttgggcacc 660
cagacctaca tctgcaacgt gaatcacaag cccagcaaca ccaaggtgga caagaaagtt 720
gagcccaaat cttgtgacaa aactcacaca tgcccaccgt gcccagcacc tgaactcctg 780
gggggaccgt cagtcttcct cttcccccca aaacccaagg acaccctcat gatctcccgg 840
acccctgagg tcacatgcgt ggtggtggac gtgagccacg aagaccctga ggtcaagttc 900
aactggtacg tggacggcgt ggaggtgcat aatgccaaga caaagccgcg ggaggagcag 960
tacaacagca cgtaccgggt ggtcagcgtc ctcaccgtcc tgcaccagga ctggctgaat 1020
ggcaaggagt acaagtgcaa ggtctccaac aaagccctcc cagcccccat cgagaaaacc 1080
atctccaaag ccaaagggca gccccgagaa ccacaggtgt acaccctgcc cccatcccgg 1140
gaggagatga ccaagaacca ggtcagcctg acctgcctgg tcaaaggctt ctatcccagc 1200
gacatcgccg tggagtggga gagcaatggg cagccggaga acaactacaa gaccacgcct 1260
cccgtgctgg actccgacgg ctccttcttc ctctacagca agctcaccgt ggacaagagc 1320
aggtggcagc aggggaacgt cttctcatgc tccgtgatgc atgaggctct gcacaaccac 1380
tacacgcaga agagcctctc cctgtctccc gggaaatccg ggggtggcgg atccgatgag 1440
gctgcaggga taggcccaga agttcctgat gaccgcgact tcgagccctc cctaggccca 1500
gtgtgcccct tccgctgtca atgccatctt cgagtggtcc agtgttctga tttgggtctg 1560
gacaaagtgc caaaggatct tccccctgac acaactctgc tagacctgca aaacaacaaa 1620
ataaccgaaa tcaaagatgg agactttaag aacctgaaga accttcacgc attgattctt 1680
gtcaacaata aaattagcaa agttagtcct ggagcattta cacctttggt gaagttggaa 1740
cgactttatc tgtccaagaa tcagctgaag gaattgccag aaaaaatgcc caaaactctt 1800
caggagctgc gtgcccatga gaatgagatc accaaagtgc gaaaagttac tttcaatgga 1860
ctgaaccaga tgattgtcat agaactgggc accaatccgc tgaagagctc aggaattgaa 1920
aatggggctt tccagggaat gaagaagctc tcctacatcc gcattgctga taccaatatc 1980
accagcattc ctcaaggtct tcctccttcc cttacggaat tacatcttga tggcaacaaa 2040
atcagcagag ttgatgcagc tagcctgaaa ggactgaata atttggctaa gttgggattg 2100
agtttcaaca gcatctctgc tgttgacaat ggctctctgg ccaacacgcc tcatctgagg 2160
gagcttcact tggacaacaa caagcttacc agagtacctg gtgggctggc agagcataag 2220
tacatccagg ttgtctacct tcataacaac aatatctctg tagttggatc aagtgacttc 2280
tgcccacctg gacacaacac caaaaaggct tcttattcgg gtgtgagtct tttcagcaac 2340
ccggtccagt actgggagat acagccatcc accttcagat gtgtctacgt gcgctctgcc 2400
attcaactcg gaaactataa gtga 2424
<210> 2
<211> 807
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic
<400> 2
Met Met Ser Phe Val Ser Leu Leu Leu Val Gly Ile Leu Phe His Ala
1 5 10 15
Thr Gln Ala Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln
20 25 30
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Tyr Thr Phe
35 40 45
Thr Asn Tyr Gly Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
Glu Trp Val Gly Trp Ile Asn Thr Tyr Thr Gly Glu Pro Thr Tyr Ala
65 70 75 80
Ala Asp Phe Lys Arg Arg Phe Thr Phe Ser Leu Asp Thr Ser Lys Ser
85 90 95
Thr Ala Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Ala Lys Tyr Pro His Tyr Tyr Gly Ser Ser His Trp Tyr
115 120 125
Phe Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser
130 135 140
Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr
145 150 155 160
Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro
165 170 175
Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val
180 185 190
His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser
195 200 205
Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile
210 215 220
Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val
225 230 235 240
Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala
245 250 255
Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro
260 265 270
Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val
275 280 285
Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val
290 295 300
Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln
305 310 315 320
Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln
325 330 335
Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala
340 345 350
Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro
355 360 365
Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr
370 375 380
Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser
385 390 395 400
Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr
405 410 415
Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr
420 425 430
Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe
435 440 445
Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys
450 455 460
Ser Leu Ser Leu Ser Pro Gly Lys Ser Gly Gly Gly Gly Ser Asp Glu
465 470 475 480
Ala Ala Gly Ile Gly Pro Glu Val Pro Asp Asp Arg Asp Phe Glu Pro
485 490 495
Ser Leu Gly Pro Val Cys Pro Phe Arg Cys Gln Cys His Leu Arg Val
500 505 510
Val Gln Cys Ser Asp Leu Gly Leu Asp Lys Val Pro Lys Asp Leu Pro
515 520 525
Pro Asp Thr Thr Leu Leu Asp Leu Gln Asn Asn Lys Ile Thr Glu Ile
530 535 540
Lys Asp Gly Asp Phe Lys Asn Leu Lys Asn Leu His Ala Leu Ile Leu
545 550 555 560
Val Asn Asn Lys Ile Ser Lys Val Ser Pro Gly Ala Phe Thr Pro Leu
565 570 575
Val Lys Leu Glu Arg Leu Tyr Leu Ser Lys Asn Gln Leu Lys Glu Leu
580 585 590
Pro Glu Lys Met Pro Lys Thr Leu Gln Glu Leu Arg Ala His Glu Asn
595 600 605
Glu Ile Thr Lys Val Arg Lys Val Thr Phe Asn Gly Leu Asn Gln Met
610 615 620
Ile Val Ile Glu Leu Gly Thr Asn Pro Leu Lys Ser Ser Gly Ile Glu
625 630 635 640
Asn Gly Ala Phe Gln Gly Met Lys Lys Leu Ser Tyr Ile Arg Ile Ala
645 650 655
Asp Thr Asn Ile Thr Ser Ile Pro Gln Gly Leu Pro Pro Ser Leu Thr
660 665 670
Glu Leu His Leu Asp Gly Asn Lys Ile Ser Arg Val Asp Ala Ala Ser
675 680 685
Leu Lys Gly Leu Asn Asn Leu Ala Lys Leu Gly Leu Ser Phe Asn Ser
690 695 700
Ile Ser Ala Val Asp Asn Gly Ser Leu Ala Asn Thr Pro His Leu Arg
705 710 715 720
Glu Leu His Leu Asp Asn Asn Lys Leu Thr Arg Val Pro Gly Gly Leu
725 730 735
Ala Glu His Lys Tyr Ile Gln Val Val Tyr Leu His Asn Asn Asn Ile
740 745 750
Ser Val Val Gly Ser Ser Asp Phe Cys Pro Pro Gly His Asn Thr Lys
755 760 765
Lys Ala Ser Tyr Ser Gly Val Ser Leu Phe Ser Asn Pro Val Gln Tyr
770 775 780
Trp Glu Ile Gln Pro Ser Thr Phe Arg Cys Val Tyr Val Arg Ser Ala
785 790 795 800
Ile Gln Leu Gly Asn Tyr Lys
805
<210> 3
<211> 702
<212> DNA
<213> Artificial Sequence
<220>
<223> synthetic
<400> 3
atgatgtcct ttgtctctct gctcctggtt ggcatcctgt tccatgccac ccaggccgac 60
atccagatga cccagtctcc atcctccctg tctgcatctg taggagacag agtcaccatc 120
acttgcagtg caagtcagga cattagcaat tatttaaact ggtatcagca gaaaccaggg 180
aaagctccta aggtcctgat ctatttcaca tccagtttgc actcaggggt cccatctagg 240
ttcagtggca gtggatctgg gacagatttc actctcacca tcagcagcct gcagcctgaa 300
gattttgcaa cttattactg ccaacagtat agtaccgtgc cttggacgtt cggccaaggg 360
accaaggtgg aaatcaaacg aactgtggct gcaccatctg tcttcatctt cccgccatct 420
gatgagcagc ttaagtccgg aactgctagc gttgtgtgcc tgctgaataa cttctatccc 480
agagaggcca aagtacagtg gaaggtggat aacgccctcc aatcgggtaa ctcccaggag 540
agtgtcacag agcaggacag caaggacagc acctacagcc tcagcagcac cctgacgctg 600
agcaaagcag actacgagaa acacaaagtc tacgcctgcg aagtcaccca tcagggcctg 660
agctcgcccg tcacaaagag cttcaacagg ggagagtgtt ag 702
<210> 4
<211> 233
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic
<400> 4
Met Met Ser Phe Val Ser Leu Leu Leu Val Gly Ile Leu Phe His Ala
1 5 10 15
Thr Gln Ala Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala
20 25 30
Ser Val Gly Asp Arg Val Thr Ile Thr Cys Ser Ala Ser Gln Asp Ile
35 40 45
Ser Asn Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys
50 55 60
Val Leu Ile Tyr Phe Thr Ser Ser Leu His Ser Gly Val Pro Ser Arg
65 70 75 80
Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser
85 90 95
Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Ser Thr
100 105 110
Val Pro Trp Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr
115 120 125
Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu
130 135 140
Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro
145 150 155 160
Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly
165 170 175
Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr
180 185 190
Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His
195 200 205
Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val
210 215 220
Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230
<210> 5
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic
<400> 5
Met Met Ser Phe Val Ser Leu Leu Leu Val Gly Ile Leu Phe His Ala
1 5 10 15
Thr Gln Ala
<210> 6
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic
<400> 6
Ser Gly Gly Gly Gly Ser
1 5
<210> 7
<211> 329
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic
<400> 7
Asp Glu Ala Ala Gly Ile Gly Pro Glu Val Pro Asp Asp Arg Asp Phe
1 5 10 15
Glu Pro Ser Leu Gly Pro Val Cys Pro Phe Arg Cys Gln Cys His Leu
20 25 30
Arg Val Val Gln Cys Ser Asp Leu Gly Leu Asp Lys Val Pro Lys Asp
35 40 45
Leu Pro Pro Asp Thr Thr Leu Leu Asp Leu Gln Asn Asn Lys Ile Thr
50 55 60
Glu Ile Lys Asp Gly Asp Phe Lys Asn Leu Lys Asn Leu His Ala Leu
65 70 75 80
Ile Leu Val Asn Asn Lys Ile Ser Lys Val Ser Pro Gly Ala Phe Thr
85 90 95
Pro Leu Val Lys Leu Glu Arg Leu Tyr Leu Ser Lys Asn Gln Leu Lys
100 105 110
Glu Leu Pro Glu Lys Met Pro Lys Thr Leu Gln Glu Leu Arg Ala His
115 120 125
Glu Asn Glu Ile Thr Lys Val Arg Lys Val Thr Phe Asn Gly Leu Asn
130 135 140
Gln Met Ile Val Ile Glu Leu Gly Thr Asn Pro Leu Lys Ser Ser Gly
145 150 155 160
Ile Glu Asn Gly Ala Phe Gln Gly Met Lys Lys Leu Ser Tyr Ile Arg
165 170 175
Ile Ala Asp Thr Asn Ile Thr Ser Ile Pro Gln Gly Leu Pro Pro Ser
180 185 190
Leu Thr Glu Leu His Leu Asp Gly Asn Lys Ile Ser Arg Val Asp Ala
195 200 205
Ala Ser Leu Lys Gly Leu Asn Asn Leu Ala Lys Leu Gly Leu Ser Phe
210 215 220
Asn Ser Ile Ser Ala Val Asp Asn Gly Ser Leu Ala Asn Thr Pro His
225 230 235 240
Leu Arg Glu Leu His Leu Asp Asn Asn Lys Leu Thr Arg Val Pro Gly
245 250 255
Gly Leu Ala Glu His Lys Tyr Ile Gln Val Val Tyr Leu His Asn Asn
260 265 270
Asn Ile Ser Val Val Gly Ser Ser Asp Phe Cys Pro Pro Gly His Asn
275 280 285
Thr Lys Lys Ala Ser Tyr Ser Gly Val Ser Leu Phe Ser Asn Pro Val
290 295 300
Gln Tyr Trp Glu Ile Gln Pro Ser Thr Phe Arg Cys Val Tyr Val Arg
305 310 315 320
Ser Ala Ile Gln Leu Gly Asn Tyr Lys
325
<210> 8
<211> 453
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic
<400> 8
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Tyr Thr Phe Thr Asn Tyr
20 25 30
Gly Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Trp Ile Asn Thr Tyr Thr Gly Glu Pro Thr Tyr Ala Ala Asp Phe
50 55 60
Lys Arg Arg Phe Thr Phe Ser Leu Asp Thr Ser Lys Ser Thr Ala Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Tyr Pro His Tyr Tyr Gly Ser Ser His Trp Tyr Phe Asp Val
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly
115 120 125
Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly
130 135 140
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
145 150 155 160
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
165 170 175
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
180 185 190
Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val
195 200 205
Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys
210 215 220
Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu
225 230 235 240
Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
245 250 255
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
260 265 270
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
275 280 285
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
290 295 300
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
305 310 315 320
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala
325 330 335
Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
340 345 350
Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln
355 360 365
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
370 375 380
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
385 390 395 400
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
405 410 415
Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser
420 425 430
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
435 440 445
Leu Ser Pro Gly Lys
450
<210> 9
<211> 214
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic
<400> 9
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Ser Ala Ser Gln Asp Ile Ser Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Val Leu Ile
35 40 45
Tyr Phe Thr Ser Ser Leu His Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Ser Thr Val Pro Trp
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 10
<211> 2409
<212> DNA
<213> Artificial Sequence
<220>
<223> synthetic
<400> 10
atgatgtcct ttgtctctct gctcctggtt ggcatcctat tccatgccac ccaggcccag 60
gtgcagctgg tggagtccgg cggcggcgtc gtgcagcccg gccggtccct gcggctgtcc 120
tgcgccgcct ccggcttcac cttctcctcc tacaccatgc actgggtgcg gcaggccccc 180
ggcaagggcc tggagtgggt gactttcatc tcctacgacg gcaacaacaa gtactacgcc 240
gactccgtga agggccggtt caccatctcc cgcgacaact ccaagaacac cctgtacctg 300
cagatgaact ccctgcgggc cgaggacacc gccatctact actgcgcccg gaccggctgg 360
ctgggcccct tcgactactg gggccagggc accctggtga ccgtgtcctc cgcctccacc 420
aagggcccat cggtcttccc cctggcaccc tctagcaaga gcacctctgg gggcacagcg 480
gccctgggct gcctggtcaa ggactacttc cccgaaccgg tgacggtgtc gtggaactca 540
ggcgccctga ccagcggcgt gcacaccttc ccggctgtcc tacagtcctc aggactctac 600
tccctcagca gcgtggtgac cgtgccctcc agcagcttgg gcacccagac ctacatctgc 660
aacgtgaatc acaagcccag caacaccaag gtggacaagc gggttgagcc caaatcttgt 720
gacaaaactc acacatgccc accgtgccca gcacctgaac tcctgggggg accgtcagtc 780
ttcctcttcc ccccaaaacc caaggacacc ctcatgatct cccggacccc tgaggtcaca 840
tgcgtggtgg tggacgtgag ccacgaagac cctgaggtca agttcaactg gtacgtggac 900
ggcgtggagg tgcataatgc caagacaaag ccgcgggagg agcagtacaa cagcacgtac 960
cgtgtggtca gcgtcctcac cgtcctgcac caggactggc tgaatggcaa ggagtacaag 1020
tgcaaggtct ccaacaaagc cctcccagcc cccatcgaga aaaccatctc caaagccaaa 1080
gggcagcccc gagaaccaca ggtgtacacc ctgcctccat cccgcgatga gctgaccaag 1140
aaccaggtca gcctgacctg cctggtcaaa ggcttctatc ccagcgacat cgccgtggag 1200
tgggagagca atgggcagcc ggagaacaac tacaagacca cgcctcccgt gctggactcc 1260
gacggctcct tcttcctcta tagcaagctc accgtggaca agagcaggtg gcagcagggg 1320
aacgtcttct catgctccgt gatgcatgag gctctgcaca accactacac gcagaagagc 1380
ctctccctgt ctcctgggaa atccgggggt ggcggatccg atgaggctgc agggataggc 1440
ccagaagttc ctgatgaccg cgacttcgag ccctccctag gcccagtgtg ccccttccgc 1500
tgtcaatgcc atcttcgagt ggtccagtgt tctgatttgg gtctggacaa agtgccaaag 1560
gatcttcccc ctgacacaac tctgctagac ctgcaaaaca acaaaataac cgaaatcaaa 1620
gatggagact ttaagaacct gaagaacctt cacgcattga ttcttgtcaa caataaaatt 1680
agcaaagtta gtcctggagc atttacacct ttggtgaagt tggaacgact ttatctgtcc 1740
aagaatcagc tgaaggaatt gccagaaaaa atgcccaaaa ctcttcagga gctgcgtgcc 1800
catgagaatg agatcaccaa agtgcgaaaa gttactttca atggactgaa ccagatgatt 1860
gtcatagaac tgggcaccaa tccgctgaag agctcaggaa ttgaaaatgg ggctttccag 1920
ggaatgaaga agctctccta catccgcatt gctgatacca atatcaccag cattcctcaa 1980
ggtcttcctc cttcccttac ggaattacat cttgatggca acaaaatcag cagagttgat 2040
gcagctagcc tgaaaggact gaataatttg gctaagttgg gattgagttt caacagcatc 2100
tctgctgttg acaatggctc tctggccaac acgcctcatc tgagggagct tcacttggac 2160
aacaacaagc ttaccagagt acctggtggg ctggcagagc ataagtacat ccaggttgtc 2220
taccttcata acaacaatat ctctgtagtt ggatcaagtg acttctgccc acctggacac 2280
aacaccaaaa aggcttctta ttcgggtgtg agtcttttca gcaacccggt ccagtactgg 2340
gagatacagc catccacctt cagatgtgtc tacgtgcgct ctgccattca actcggaaac 2400
tataagtga 2409
<210> 11
<211> 802
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic
<400> 11
Met Met Ser Phe Val Ser Leu Leu Leu Val Gly Ile Leu Phe His Ala
1 5 10 15
Thr Gln Ala Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln
20 25 30
Pro Gly Arg Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
35 40 45
Ser Ser Tyr Thr Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
Glu Trp Val Thr Phe Ile Ser Tyr Asp Gly Asn Asn Lys Tyr Tyr Ala
65 70 75 80
Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn
85 90 95
Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Ile
100 105 110
Tyr Tyr Cys Ala Arg Thr Gly Trp Leu Gly Pro Phe Asp Tyr Trp Gly
115 120 125
Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
130 135 140
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
145 150 155 160
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
165 170 175
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
180 185 190
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
195 200 205
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
210 215 220
Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys
225 230 235 240
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly
245 250 255
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
260 265 270
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
275 280 285
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
290 295 300
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
305 310 315 320
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
325 330 335
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile
340 345 350
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
355 360 365
Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser
370 375 380
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
385 390 395 400
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
405 410 415
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
420 425 430
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
435 440 445
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
450 455 460
Pro Gly Lys Ser Gly Gly Gly Gly Ser Asp Glu Ala Ala Gly Ile Gly
465 470 475 480
Pro Glu Val Pro Asp Asp Arg Asp Phe Glu Pro Ser Leu Gly Pro Val
485 490 495
Cys Pro Phe Arg Cys Gln Cys His Leu Arg Val Val Gln Cys Ser Asp
500 505 510
Leu Gly Leu Asp Lys Val Pro Lys Asp Leu Pro Pro Asp Thr Thr Leu
515 520 525
Leu Asp Leu Gln Asn Asn Lys Ile Thr Glu Ile Lys Asp Gly Asp Phe
530 535 540
Lys Asn Leu Lys Asn Leu His Ala Leu Ile Leu Val Asn Asn Lys Ile
545 550 555 560
Ser Lys Val Ser Pro Gly Ala Phe Thr Pro Leu Val Lys Leu Glu Arg
565 570 575
Leu Tyr Leu Ser Lys Asn Gln Leu Lys Glu Leu Pro Glu Lys Met Pro
580 585 590
Lys Thr Leu Gln Glu Leu Arg Ala His Glu Asn Glu Ile Thr Lys Val
595 600 605
Arg Lys Val Thr Phe Asn Gly Leu Asn Gln Met Ile Val Ile Glu Leu
610 615 620
Gly Thr Asn Pro Leu Lys Ser Ser Gly Ile Glu Asn Gly Ala Phe Gln
625 630 635 640
Gly Met Lys Lys Leu Ser Tyr Ile Arg Ile Ala Asp Thr Asn Ile Thr
645 650 655
Ser Ile Pro Gln Gly Leu Pro Pro Ser Leu Thr Glu Leu His Leu Asp
660 665 670
Gly Asn Lys Ile Ser Arg Val Asp Ala Ala Ser Leu Lys Gly Leu Asn
675 680 685
Asn Leu Ala Lys Leu Gly Leu Ser Phe Asn Ser Ile Ser Ala Val Asp
690 695 700
Asn Gly Ser Leu Ala Asn Thr Pro His Leu Arg Glu Leu His Leu Asp
705 710 715 720
Asn Asn Lys Leu Thr Arg Val Pro Gly Gly Leu Ala Glu His Lys Tyr
725 730 735
Ile Gln Val Val Tyr Leu His Asn Asn Asn Ile Ser Val Val Gly Ser
740 745 750
Ser Asp Phe Cys Pro Pro Gly His Asn Thr Lys Lys Ala Ser Tyr Ser
755 760 765
Gly Val Ser Leu Phe Ser Asn Pro Val Gln Tyr Trp Glu Ile Gln Pro
770 775 780
Ser Thr Phe Arg Cys Val Tyr Val Arg Ser Ala Ile Gln Leu Gly Asn
785 790 795 800
Tyr Lys
<210> 12
<211> 705
<212> DNA
<213> Artificial Sequence
<220>
<223> synthetic
<400> 12
atgatgtcct ttgtctctct gctcctggtt ggcatcctat tccatgccac ccaggccgag 60
atcgtgctga cccagtcccc cggcaccctg tccctgtccc ccggcgagcg ggccaccctg 120
tcctgccggg cctcccagtc cgtgggctcc tcctacctgg cctggtacca gcagaagccc 180
ggccaggccc cccggctgct gatctacggc gccttctccc gcgccaccgg catccccgac 240
cggttctccg gctccggctc cggcaccgac ttcaccctga ccatctcccg gctggagccc 300
gaggacttcg ccgtgtacta ctgccagcag tacggctcct ccccctggac cttcggccag 360
ggcaccaagg tggagatcaa gcgaactgtg gctgcaccat ctgtcttcat cttcccgcca 420
tctgatgagc agcttaagtc cggaactgct agcgttgtgt gcctgctgaa taacttctat 480
cccagagagg ccaaagtaca gtggaaggtg gataacgccc tccaatcggg taactcccag 540
gagagtgtca cagagcagga cagcaaggac agcacctaca gcctcagcag caccctgacg 600
ctgagcaaag cagactacga gaaacacaaa gtctacgcct gcgaagtcac ccatcagggc 660
ctgagctcgc ccgtcacaaa gagcttcaac aggggagagt gttag 705
<210> 13
<211> 234
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic
<400> 13
Met Met Ser Phe Val Ser Leu Leu Leu Val Gly Ile Leu Phe His Ala
1 5 10 15
Thr Gln Ala Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu
20 25 30
Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val
35 40 45
Gly Ser Ser Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro
50 55 60
Arg Leu Leu Ile Tyr Gly Ala Phe Ser Arg Ala Thr Gly Ile Pro Asp
65 70 75 80
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
85 90 95
Arg Leu Glu Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly
100 105 110
Ser Ser Pro Trp Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg
115 120 125
Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln
130 135 140
Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr
145 150 155 160
Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser
165 170 175
Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr
180 185 190
Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys
195 200 205
His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro
210 215 220
Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230
<210> 14
<211> 448
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic
<400> 14
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Thr Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Thr Phe Ile Ser Tyr Asp Gly Asn Asn Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Ile Tyr Tyr Cys
85 90 95
Ala Arg Thr Gly Trp Leu Gly Pro Phe Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys Asp Lys Thr
210 215 220
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser
225 230 235 240
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
245 250 255
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
260 265 270
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
275 280 285
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val
290 295 300
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
305 310 315 320
Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr
325 330 335
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
340 345 350
Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys
355 360 365
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
370 375 380
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
385 390 395 400
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser
405 410 415
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
420 425 430
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 15
<211> 215
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic
<400> 15
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Gly Ser Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Phe Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Ser Ser Pro
85 90 95
Trp Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala
100 105 110
Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser
115 120 125
Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu
130 135 140
Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser
145 150 155 160
Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu
165 170 175
Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val
180 185 190
Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys
195 200 205
Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 16
<211> 2409
<212> DNA
<213> Artificial Sequence
<220>
<223> synthetic
<400> 16
atgatgtcct ttgtctctct gctcctggtt ggcatcctat tccatgccac ccaggccgag 60
gtacaactcg tggaatccgg cgggggactc gtccaaccgg gtggtagtct caggttgagc 120
tgcgctgcga gcggtttcac tttctcagac tcatggattc attgggtgcg ccaagcacct 180
ggaaaagggt tggaatgggt tgcctggatc tctccttacg gaggttctac ctactacgct 240
gattcagtaa aggggcggtt cacaatttca gccgatactt ccaaaaatac ggcttacctg 300
caaatgaact ctttgagggc ggaggatacg gcggtctact actgtgcacg caggcactgg 360
cctggagggt tcgattattg gggtcaaggc actttggtaa ccgtatcctc cgcttctacc 420
aaaggcccat cagtatttcc tttggctccc agctctaagt ccacttccgg tggaactgcc 480
gcacttggat gtctcgtcaa agactacttt cctgagccgg taactgtgtc atggaactcc 540
ggcgccctca ctagcggcgt ccatacattt ccagcggttc tccagtcaag tggcctctac 600
agcctgtcca gtgtagttac tgtcccgtct tctagtctgg gaacgcaaac atatatttgc 660
aatgtgaatc ataagcctag taacacaaaa gtcgataaaa aagtggagcc gaaaagttgt 720
gacaaaacgc atacctgtcc gccttgtccg gcccccgaac tcttgggcgg cccatcagtc 780
tttctcttcc cgcccaaacc taaggacacg ttgatgataa gtcgcacgcc cgaggttaca 840
tgcgtcgtag tcgatgtcag ccacgaggat ccggaggtaa agtttaactg gtatgtagac 900
ggagttgaag tacacaacgc caaaactaaa ccgagagagg agcagtacgc atcaacctat 960
cgcgtagtat ctgtattgac ggtccttcac caagactggc tcaatgggaa agaatacaag 1020
tgcaaagttt ctaataaagc cctccctgca ccaatcgaaa agactatttc aaaagccaaa 1080
ggacaaccaa gagaaccaca agtttataca ttgccaccta gtcgcgagga gatgactaaa 1140
aaccaagtgt cccttacttg tctcgtaaag ggtttctatc caagcgacat agcagttgag 1200
tgggaaagta atggccagcc ggaaaacaac tacaagacga cccccccggt tctcgactcc 1260
gatggatcat tctttttgta tagtaaactc acagttgata agagtcgatg gcagcagggg 1320
aatgtttttt cttgctctgt gatgcacgag gcgctccaca accactatac gcaaaagtcc 1380
ctcagcctga gccccgggaa atccgggggt ggcggatccg atgaggctgc agggataggc 1440
ccagaagttc ctgatgaccg cgacttcgag ccctccctag gcccagtgtg ccccttccgc 1500
tgtcaatgcc atcttcgagt ggtccagtgt tctgatttgg gtctggacaa agtgccaaag 1560
gatcttcccc ctgacacaac tctgctagac ctgcaaaaca acaaaataac cgaaatcaaa 1620
gatggagact ttaagaacct gaagaacctt cacgcattga ttcttgtcaa caataaaatt 1680
agcaaagtta gtcctggagc atttacacct ttggtgaagt tggaacgact ttatctgtcc 1740
aagaatcagc tgaaggaatt gccagaaaaa atgcccaaaa ctcttcagga gctgcgtgcc 1800
catgagaatg agatcaccaa agtgcgaaaa gttactttca atggactgaa ccagatgatt 1860
gtcatagaac tgggcaccaa tccgctgaag agctcaggaa ttgaaaatgg ggctttccag 1920
ggaatgaaga agctctccta catccgcatt gctgatacca atatcaccag cattcctcaa 1980
ggtcttcctc cttcccttac ggaattacat cttgatggca acaaaatcag cagagttgat 2040
gcagctagcc tgaaaggact gaataatttg gctaagttgg gattgagttt caacagcatc 2100
tctgctgttg acaatggctc tctggccaac acgcctcatc tgagggagct tcacttggac 2160
aacaacaagc ttaccagagt acctggtggg ctggcagagc ataagtacat ccaggttgtc 2220
taccttcata acaacaatat ctctgtagtt ggatcaagtg acttctgccc acctggacac 2280
aacaccaaaa aggcttctta ttcgggtgtg agtcttttca gcaacccggt ccagtactgg 2340
gagatacagc catccacctt cagatgtgtc tacgtgcgct ctgccattca actcggaaac 2400
tataagtga 2409
<210> 17
<211> 802
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic
<400> 17
Met Met Ser Phe Val Ser Leu Leu Leu Val Gly Ile Leu Phe His Ala
1 5 10 15
Thr Gln Ala Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln
20 25 30
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
35 40 45
Ser Asp Ser Trp Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
Glu Trp Val Ala Trp Ile Ser Pro Tyr Gly Gly Ser Thr Tyr Tyr Ala
65 70 75 80
Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Ala Asp Thr Ser Lys Asn
85 90 95
Thr Ala Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Ala Arg Arg His Trp Pro Gly Gly Phe Asp Tyr Trp Gly
115 120 125
Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
130 135 140
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
145 150 155 160
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
165 170 175
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
180 185 190
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
195 200 205
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
210 215 220
Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
225 230 235 240
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly
245 250 255
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
260 265 270
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
275 280 285
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
290 295 300
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Ala Ser Thr Tyr
305 310 315 320
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
325 330 335
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile
340 345 350
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
355 360 365
Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser
370 375 380
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
385 390 395 400
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
405 410 415
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
420 425 430
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
435 440 445
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
450 455 460
Pro Gly Lys Ser Gly Gly Gly Gly Ser Asp Glu Ala Ala Gly Ile Gly
465 470 475 480
Pro Glu Val Pro Asp Asp Arg Asp Phe Glu Pro Ser Leu Gly Pro Val
485 490 495
Cys Pro Phe Arg Cys Gln Cys His Leu Arg Val Val Gln Cys Ser Asp
500 505 510
Leu Gly Leu Asp Lys Val Pro Lys Asp Leu Pro Pro Asp Thr Thr Leu
515 520 525
Leu Asp Leu Gln Asn Asn Lys Ile Thr Glu Ile Lys Asp Gly Asp Phe
530 535 540
Lys Asn Leu Lys Asn Leu His Ala Leu Ile Leu Val Asn Asn Lys Ile
545 550 555 560
Ser Lys Val Ser Pro Gly Ala Phe Thr Pro Leu Val Lys Leu Glu Arg
565 570 575
Leu Tyr Leu Ser Lys Asn Gln Leu Lys Glu Leu Pro Glu Lys Met Pro
580 585 590
Lys Thr Leu Gln Glu Leu Arg Ala His Glu Asn Glu Ile Thr Lys Val
595 600 605
Arg Lys Val Thr Phe Asn Gly Leu Asn Gln Met Ile Val Ile Glu Leu
610 615 620
Gly Thr Asn Pro Leu Lys Ser Ser Gly Ile Glu Asn Gly Ala Phe Gln
625 630 635 640
Gly Met Lys Lys Leu Ser Tyr Ile Arg Ile Ala Asp Thr Asn Ile Thr
645 650 655
Ser Ile Pro Gln Gly Leu Pro Pro Ser Leu Thr Glu Leu His Leu Asp
660 665 670
Gly Asn Lys Ile Ser Arg Val Asp Ala Ala Ser Leu Lys Gly Leu Asn
675 680 685
Asn Leu Ala Lys Leu Gly Leu Ser Phe Asn Ser Ile Ser Ala Val Asp
690 695 700
Asn Gly Ser Leu Ala Asn Thr Pro His Leu Arg Glu Leu His Leu Asp
705 710 715 720
Asn Asn Lys Leu Thr Arg Val Pro Gly Gly Leu Ala Glu His Lys Tyr
725 730 735
Ile Gln Val Val Tyr Leu His Asn Asn Asn Ile Ser Val Val Gly Ser
740 745 750
Ser Asp Phe Cys Pro Pro Gly His Asn Thr Lys Lys Ala Ser Tyr Ser
755 760 765
Gly Val Ser Leu Phe Ser Asn Pro Val Gln Tyr Trp Glu Ile Gln Pro
770 775 780
Ser Thr Phe Arg Cys Val Tyr Val Arg Ser Ala Ile Gln Leu Gly Asn
785 790 795 800
Tyr Lys
<210> 18
<211> 702
<212> DNA
<213> Artificial Sequence
<220>
<223> synthetic
<400> 18
atgatgtcct ttgtctctct gctcctggtt ggcatcctat tccatgccac ccaggccgac 60
attcagatga cacaatcacc tagcagtctg agtgcgagcg taggtgatcg cgtaacgatt 120
acctgcaggg cctctcaaga cgtgtcaacg gcagtggcgt ggtaccagca gaagcctggt 180
aaagctccta agctcctcat ctattcagct tccttcttgt atagtggagt accgtcaaga 240
ttttccggaa gcggatcagg tacagatttt actttgacta tcagtagttt gcagccagag 300
gatttcgcta catattactg tcaacaatat ctctatcacc ctgccacttt tggacaaggg 360
actaaagtcg aaataaaacg aacagtggcc gcaccaagcg tttttatctt tcccccatcc 420
gacgagcagt tgaagagcgg caccgcgtcc gtggtctgcc tgttgaataa tttctatcca 480
agggaggcaa aagtgcaatg gaaagttgat aatgcgcttc aatccggaaa ctcacaagaa 540
tcagtaacag aacaagactc taaagacagt acatattctc ttagtagcac actcactctt 600
tcaaaggctg actatgagaa acataaagtg tacgcttgtg aagtgacaca tcaaggtctt 660
agctccccag taactaagag ctttaatagg ggcgagtgct ga 702
<210> 19
<211> 233
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic
<400> 19
Met Met Ser Phe Val Ser Leu Leu Leu Val Gly Ile Leu Phe His Ala
1 5 10 15
Thr Gln Ala Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala
20 25 30
Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Asp Val
35 40 45
Ser Thr Ala Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys
50 55 60
Leu Leu Ile Tyr Ser Ala Ser Phe Leu Tyr Ser Gly Val Pro Ser Arg
65 70 75 80
Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser
85 90 95
Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Leu Tyr
100 105 110
His Pro Ala Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr
115 120 125
Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu
130 135 140
Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro
145 150 155 160
Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly
165 170 175
Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr
180 185 190
Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His
195 200 205
Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val
210 215 220
Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230
<210> 20
<211> 448
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic
<400> 20
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Ser
20 25 30
Trp Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Trp Ile Ser Pro Tyr Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Ala Asp Thr Ser Lys Asn Thr Ala Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Arg His Trp Pro Gly Gly Phe Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
210 215 220
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser
225 230 235 240
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
245 250 255
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
260 265 270
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
275 280 285
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Ala Ser Thr Tyr Arg Val Val
290 295 300
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
305 310 315 320
Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr
325 330 335
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
340 345 350
Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys
355 360 365
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
370 375 380
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
385 390 395 400
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser
405 410 415
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
420 425 430
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 21
<211> 214
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic
<400> 21
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Asp Val Ser Thr Ala
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ser Ala Ser Phe Leu Tyr Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Leu Tyr His Pro Ala
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 22
<211> 2415
<212> DNA
<213> Artificial Sequence
<220>
<223> synthetic
<400> 22
atgatgtcct ttgtctctct gctcctggtt ggcatcctat tccatgccac ccaggccgag 60
gtacagcttt tggagtcagg cggggggctc gtccaacctg gggggtcact ccggttgtca 120
tgtgctgcca gtggcttcac attctcatct tacattatga tgtgggttcg acaggcccct 180
gggaaggggc tggagtgggt ttcctccatc tacccctccg ggggaattac cttctatgcc 240
gacacggtaa agggtcgctt cactataagt cgagataaca gcaaaaatac gctgtatctc 300
cagatgaact ctctcagggc tgaggacaca gctgtatatt actgcgcgcg gattaagttg 360
gggaccgtca caacagtgga ttactggggt caaggcactc tggtaaccgt atcctcagca 420
tccaccaagg gcccaagtgt attcccgctg gccccttcaa gtaaatccac gtctggcggc 480
acagccgctc tcggttgcct ggttaaggac tacttcccag aacctgtcac tgtcagttgg 540
aactcaggcg cattgacatc tggtgtccat acattccccg cagtcctgca aagctctgga 600
ctttacagtc ttagtagcgt agtgacagtc ccatcttcaa gtcttgggac ccaaacttat 660
atttgcaacg taaatcataa accctccaac actaaagtag acaaaaaagt agagccgaaa 720
tcttgcgaca aaacgcatac atgcccacca tgtcccgctc cggaactcct gggcggcccg 780
tccgtttttc tctttccccc aaagcccaag gatacgctta tgatcagcag aacaccggaa 840
gttacttgtg tagtcgttga cgtgtctcac gaagatcccg aagtcaaatt taattggtat 900
gtggatggcg tcgaagtgca caacgcaaaa accaaaccca gagaggaaca gtataacagc 960
acgtatcgag tggtctccgt acttacggtc ctccaccagg actggttgaa tggcaaggag 1020
tacaagtgca aagtgagcaa taaagcgttg ccagccccga tcgaaaaaac catcagcaag 1080
gccaagggac agcctagaga gccgcaggtt tacaccttgc cgccatcaag ggatgaactg 1140
actaaaaacc aggtatccct gacctgcctg gttaagggtt tttaccccag tgatatagcg 1200
gttgaatggg agtctaacgg gcagccagag aacaactaca aaacgacacc tcccgttctg 1260
gattccgatg gcagcttttt cttgtattct aaactcaccg tggataaatc ccgatggcag 1320
caaggcaacg tcttctcctg cagcgtgatg catgaagcct tgcacaacca ctatacccaa 1380
aagagtctca gcctgtcacc cgggaaatcc gggggtggcg gatccgatga ggctgcaggg 1440
ataggcccag aagttcctga tgaccgcgac ttcgagccct ccctaggccc agtgtgcccc 1500
ttccgctgtc aatgccatct tcgagtggtc cagtgttctg atttgggtct ggacaaagtg 1560
ccaaaggatc ttccccctga cacaactctg ctagacctgc aaaacaacaa aataaccgaa 1620
atcaaagatg gagactttaa gaacctgaag aaccttcacg cattgattct tgtcaacaat 1680
aaaattagca aagttagtcc tggagcattt acacctttgg tgaagttgga acgactttat 1740
ctgtccaaga atcagctgaa ggaattgcca gaaaaaatgc ccaaaactct tcaggagctg 1800
cgtgcccatg agaatgagat caccaaagtg cgaaaagtta ctttcaatgg actgaaccag 1860
atgattgtca tagaactggg caccaatccg ctgaagagct caggaattga aaatggggct 1920
ttccagggaa tgaagaagct ctcctacatc cgcattgctg ataccaatat caccagcatt 1980
cctcaaggtc ttcctccttc ccttacggaa ttacatcttg atggcaacaa aatcagcaga 2040
gttgatgcag ctagcctgaa aggactgaat aatttggcta agttgggatt gagtttcaac 2100
agcatctctg ctgttgacaa tggctctctg gccaacacgc ctcatctgag ggagcttcac 2160
ttggacaaca acaagcttac cagagtacct ggtgggctgg cagagcataa gtacatccag 2220
gttgtctacc ttcataacaa caatatctct gtagttggat caagtgactt ctgcccacct 2280
ggacacaaca ccaaaaaggc ttcttattcg ggtgtgagtc ttttcagcaa cccggtccag 2340
tactgggaga tacagccatc caccttcaga tgtgtctacg tgcgctctgc cattcaactc 2400
ggaaactata agtga 2415
<210> 23
<211> 804
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic
<400> 23
Met Met Ser Phe Val Ser Leu Leu Leu Val Gly Ile Leu Phe His Ala
1 5 10 15
Thr Gln Ala Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln
20 25 30
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
35 40 45
Ser Ser Tyr Ile Met Met Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
Glu Trp Val Ser Ser Ile Tyr Pro Ser Gly Gly Ile Thr Phe Tyr Ala
65 70 75 80
Asp Thr Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn
85 90 95
Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Ala Arg Ile Lys Leu Gly Thr Val Thr Thr Val Asp Tyr
115 120 125
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly
130 135 140
Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly
145 150 155 160
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
165 170 175
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
180 185 190
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
195 200 205
Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val
210 215 220
Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys
225 230 235 240
Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu
245 250 255
Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
260 265 270
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
275 280 285
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
290 295 300
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
305 310 315 320
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
325 330 335
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala
340 345 350
Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
355 360 365
Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln
370 375 380
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
385 390 395 400
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
405 410 415
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
420 425 430
Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser
435 440 445
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
450 455 460
Leu Ser Pro Gly Lys Ser Gly Gly Gly Gly Ser Asp Glu Ala Ala Gly
465 470 475 480
Ile Gly Pro Glu Val Pro Asp Asp Arg Asp Phe Glu Pro Ser Leu Gly
485 490 495
Pro Val Cys Pro Phe Arg Cys Gln Cys His Leu Arg Val Val Gln Cys
500 505 510
Ser Asp Leu Gly Leu Asp Lys Val Pro Lys Asp Leu Pro Pro Asp Thr
515 520 525
Thr Leu Leu Asp Leu Gln Asn Asn Lys Ile Thr Glu Ile Lys Asp Gly
530 535 540
Asp Phe Lys Asn Leu Lys Asn Leu His Ala Leu Ile Leu Val Asn Asn
545 550 555 560
Lys Ile Ser Lys Val Ser Pro Gly Ala Phe Thr Pro Leu Val Lys Leu
565 570 575
Glu Arg Leu Tyr Leu Ser Lys Asn Gln Leu Lys Glu Leu Pro Glu Lys
580 585 590
Met Pro Lys Thr Leu Gln Glu Leu Arg Ala His Glu Asn Glu Ile Thr
595 600 605
Lys Val Arg Lys Val Thr Phe Asn Gly Leu Asn Gln Met Ile Val Ile
610 615 620
Glu Leu Gly Thr Asn Pro Leu Lys Ser Ser Gly Ile Glu Asn Gly Ala
625 630 635 640
Phe Gln Gly Met Lys Lys Leu Ser Tyr Ile Arg Ile Ala Asp Thr Asn
645 650 655
Ile Thr Ser Ile Pro Gln Gly Leu Pro Pro Ser Leu Thr Glu Leu His
660 665 670
Leu Asp Gly Asn Lys Ile Ser Arg Val Asp Ala Ala Ser Leu Lys Gly
675 680 685
Leu Asn Asn Leu Ala Lys Leu Gly Leu Ser Phe Asn Ser Ile Ser Ala
690 695 700
Val Asp Asn Gly Ser Leu Ala Asn Thr Pro His Leu Arg Glu Leu His
705 710 715 720
Leu Asp Asn Asn Lys Leu Thr Arg Val Pro Gly Gly Leu Ala Glu His
725 730 735
Lys Tyr Ile Gln Val Val Tyr Leu His Asn Asn Asn Ile Ser Val Val
740 745 750
Gly Ser Ser Asp Phe Cys Pro Pro Gly His Asn Thr Lys Lys Ala Ser
755 760 765
Tyr Ser Gly Val Ser Leu Phe Ser Asn Pro Val Gln Tyr Trp Glu Ile
770 775 780
Gln Pro Ser Thr Phe Arg Cys Val Tyr Val Arg Ser Ala Ile Gln Leu
785 790 795 800
Gly Asn Tyr Lys
<210> 24
<211> 708
<212> DNA
<213> Artificial Sequence
<220>
<223> synthetic
<400> 24
atgatgtcct ttgtctctct gctcctggtt ggcatcctat tccatgccac ccaggcccag 60
tctgcactta cacaaccggc gtccgtttcc ggatctccag gacagagcat tactatcagt 120
tgcacgggaa cctcctcaga cgtagggggg tataattatg tgtcttggta tcaacagcat 180
cccgggaaag cccccaaact gatgatctac gatgtcagca atagaccaag cggtgtgagt 240
aatcgattta gcgggtctaa atctggtaac acagcatccc tcactattag tggactgcaa 300
gcagaagatg aggcagacta ttattgcagt agctatacgt ctagttccac ccgcgttttt 360
ggcactggga cgaaagtcac cgttctcgga caaccaaaag caaaccccac cgtgactctg 420
tttccgccta gcagcgaaga attgcaggcc aataaggcga cactcgtatg ccttatctcc 480
gacttctacc cgggcgctgt gacagtcgcg tggaaagccg acggcagccc tgttaaagct 540
ggagtcgaga ccacgaagcc gtccaagcag agtaacaata agtatgctgc atccagttat 600
ctctctctca ctccggaaca gtggaagtcc catcggtcct atagttgcca agtgacccat 660
gagggttcca ccgtagagaa aacggtagca cctaccgaat gtagttga 708
<210> 25
<211> 235
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic
<400> 25
Met Met Ser Phe Val Ser Leu Leu Leu Val Gly Ile Leu Phe His Ala
1 5 10 15
Thr Gln Ala Gln Ser Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser
20 25 30
Pro Gly Gln Ser Ile Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val
35 40 45
Gly Gly Tyr Asn Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala
50 55 60
Pro Lys Leu Met Ile Tyr Asp Val Ser Asn Arg Pro Ser Gly Val Ser
65 70 75 80
Asn Arg Phe Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile
85 90 95
Ser Gly Leu Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr
100 105 110
Thr Ser Ser Ser Thr Arg Val Phe Gly Thr Gly Thr Lys Val Thr Val
115 120 125
Leu Gly Gln Pro Lys Ala Asn Pro Thr Val Thr Leu Phe Pro Pro Ser
130 135 140
Ser Glu Glu Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser
145 150 155 160
Asp Phe Tyr Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Gly Ser
165 170 175
Pro Val Lys Ala Gly Val Glu Thr Thr Lys Pro Ser Lys Gln Ser Asn
180 185 190
Asn Lys Tyr Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp
195 200 205
Lys Ser His Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr
210 215 220
Val Glu Lys Thr Val Ala Pro Thr Glu Cys Ser
225 230 235
<210> 26
<211> 450
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic
<400> 26
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ile Met Met Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ser Ile Tyr Pro Ser Gly Gly Ile Thr Phe Tyr Ala Asp Thr Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ile Lys Leu Gly Thr Val Thr Thr Val Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Lys
450
<210> 27
<211> 216
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic
<400> 27
Gln Ser Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Ile Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr
20 25 30
Asn Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu
35 40 45
Met Ile Tyr Asp Val Ser Asn Arg Pro Ser Gly Val Ser Asn Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser
85 90 95
Ser Thr Arg Val Phe Gly Thr Gly Thr Lys Val Thr Val Leu Gly Gln
100 105 110
Pro Lys Ala Asn Pro Thr Val Thr Leu Phe Pro Pro Ser Ser Glu Glu
115 120 125
Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr
130 135 140
Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Gly Ser Pro Val Lys
145 150 155 160
Ala Gly Val Glu Thr Thr Lys Pro Ser Lys Gln Ser Asn Asn Lys Tyr
165 170 175
Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His
180 185 190
Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys
195 200 205
Thr Val Ala Pro Thr Glu Cys Ser
210 215
<210> 28
<211> 2418
<212> DNA
<213> Artificial Sequence
<220>
<223> synthetic
<400> 28
atgatgtcct ttgtctctct gctcctggtt ggcatcctat tccatgccac ccaggccgag 60
gtccagcttg ttgaaagcgg tggtggcctc gtgcagcctg gtggcagttt gcggttgtct 120
tgcgcagcta gtggttttac cttctccaga tactggatgt catgggtccg acaggcccct 180
ggcaaggggc ttgaatgggt tgcaaatata aagcaggacg gttctgaaaa gtactatgta 240
gactccgtca aaggaagatt tactattagt cgagacaacg ccaagaatag cctctacctt 300
cagatgaatt ctttgcgagc ggaggacaca gccgtatatt actgcgcacg agaagggggg 360
tggttcggtg aactggcttt tgactactgg gggcaaggta cgcttgtcac ggtgagctct 420
gcctctacaa aggggccgtc tgtgtttcca cttgctccat ctagtaagtc aacttctgga 480
ggtactgcgg cattgggatg ccttgttaag gattattttc ccgaacctgt aactgtgagc 540
tggaattcag gtgccctcac ctctggtgta catacctttc cagcagtttt gcaatcttcc 600
ggtttgtact ctcttagttc agttgtaact gtcccctctt cctctcttgg tacccaaaca 660
tacatttgta atgtcaatca caaaccaagc aataccaagg tagacaagcg ggtggaaccc 720
aaatcttgtg acaaaactca tacctgccca ccatgtcccg ccccggagtt tgaaggaggt 780
ccaagtgtat ttcttttccc gcctaagcct aaggataccc tcatgataag tcggacacca 840
gaggtgacgt gtgttgtggt agacgtgagt cacgaagatc ccgaagttaa atttaattgg 900
tatgtggacg gggtggaagt ccataacgcg aagacaaagc cacgcgaaga gcagtacaat 960
tccacgtaca gggtggttag cgtgcttacc gtcctgcatc aagattggct gaacgggaaa 1020
gaatacaaat gcaaagtatc caacaaggcg ttgcctgcga gtatcgagaa aacgatttct 1080
aaagctaaag gacaaccccg ggaaccccag gtctatacac tgccgcccag cagagaagag 1140
atgacgaaaa atcaagtatc ccttacgtgt ctcgtcaaag gcttctaccc ttccgatatt 1200
gctgtagagt gggaatctaa cgggcagccg gaaaataact acaagactac tccgccagta 1260
cttgattcag acggctcctt cttcctttat tcaaaactca ccgtagataa aagtaggtgg 1320
caacaaggta atgtttttag ctgtagcgta atgcacgaag cgttgcataa ccattataca 1380
cagaaatcac tcagcctgtc ccccgggaaa tccgggggtg gcggatccga tgaggctgca 1440
gggataggcc cagaagttcc tgatgaccgc gacttcgagc cctccctagg cccagtgtgc 1500
cccttccgct gtcaatgcca tcttcgagtg gtccagtgtt ctgatttggg tctggacaaa 1560
gtgccaaagg atcttccccc tgacacaact ctgctagacc tgcaaaacaa caaaataacc 1620
gaaatcaaag atggagactt taagaacctg aagaaccttc acgcattgat tcttgtcaac 1680
aataaaatta gcaaagttag tcctggagca tttacacctt tggtgaagtt ggaacgactt 1740
tatctgtcca agaatcagct gaaggaattg ccagaaaaaa tgcccaaaac tcttcaggag 1800
ctgcgtgccc atgagaatga gatcaccaaa gtgcgaaaag ttactttcaa tggactgaac 1860
cagatgattg tcatagaact gggcaccaat ccgctgaaga gctcaggaat tgaaaatggg 1920
gctttccagg gaatgaagaa gctctcctac atccgcattg ctgataccaa tatcaccagc 1980
attcctcaag gtcttcctcc ttcccttacg gaattacatc ttgatggcaa caaaatcagc 2040
agagttgatg cagctagcct gaaaggactg aataatttgg ctaagttggg attgagtttc 2100
aacagcatct ctgctgttga caatggctct ctggccaaca cgcctcatct gagggagctt 2160
cacttggaca acaacaagct taccagagta cctggtgggc tggcagagca taagtacatc 2220
caggttgtct accttcataa caacaatatc tctgtagttg gatcaagtga cttctgccca 2280
cctggacaca acaccaaaaa ggcttcttat tcgggtgtga gtcttttcag caacccggtc 2340
cagtactggg agatacagcc atccaccttc agatgtgtct acgtgcgctc tgccattcaa 2400
ctcggaaact ataagtga 2418
<210> 29
<211> 805
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic
<400> 29
Met Met Ser Phe Val Ser Leu Leu Leu Val Gly Ile Leu Phe His Ala
1 5 10 15
Thr Gln Ala Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln
20 25 30
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
35 40 45
Ser Arg Tyr Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
Glu Trp Val Ala Asn Ile Lys Gln Asp Gly Ser Glu Lys Tyr Tyr Val
65 70 75 80
Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn
85 90 95
Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Ala Arg Glu Gly Gly Trp Phe Gly Glu Leu Ala Phe Asp
115 120 125
Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys
130 135 140
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
145 150 155 160
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
165 170 175
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
180 185 190
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
195 200 205
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
210 215 220
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro
225 230 235 240
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
245 250 255
Phe Glu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
260 265 270
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
275 280 285
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
290 295 300
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
305 310 315 320
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
325 330 335
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
340 345 350
Ala Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
355 360 365
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn
370 375 380
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
385 390 395 400
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
405 410 415
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
420 425 430
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
435 440 445
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
450 455 460
Ser Leu Ser Pro Gly Lys Ser Gly Gly Gly Gly Ser Asp Glu Ala Ala
465 470 475 480
Gly Ile Gly Pro Glu Val Pro Asp Asp Arg Asp Phe Glu Pro Ser Leu
485 490 495
Gly Pro Val Cys Pro Phe Arg Cys Gln Cys His Leu Arg Val Val Gln
500 505 510
Cys Ser Asp Leu Gly Leu Asp Lys Val Pro Lys Asp Leu Pro Pro Asp
515 520 525
Thr Thr Leu Leu Asp Leu Gln Asn Asn Lys Ile Thr Glu Ile Lys Asp
530 535 540
Gly Asp Phe Lys Asn Leu Lys Asn Leu His Ala Leu Ile Leu Val Asn
545 550 555 560
Asn Lys Ile Ser Lys Val Ser Pro Gly Ala Phe Thr Pro Leu Val Lys
565 570 575
Leu Glu Arg Leu Tyr Leu Ser Lys Asn Gln Leu Lys Glu Leu Pro Glu
580 585 590
Lys Met Pro Lys Thr Leu Gln Glu Leu Arg Ala His Glu Asn Glu Ile
595 600 605
Thr Lys Val Arg Lys Val Thr Phe Asn Gly Leu Asn Gln Met Ile Val
610 615 620
Ile Glu Leu Gly Thr Asn Pro Leu Lys Ser Ser Gly Ile Glu Asn Gly
625 630 635 640
Ala Phe Gln Gly Met Lys Lys Leu Ser Tyr Ile Arg Ile Ala Asp Thr
645 650 655
Asn Ile Thr Ser Ile Pro Gln Gly Leu Pro Pro Ser Leu Thr Glu Leu
660 665 670
His Leu Asp Gly Asn Lys Ile Ser Arg Val Asp Ala Ala Ser Leu Lys
675 680 685
Gly Leu Asn Asn Leu Ala Lys Leu Gly Leu Ser Phe Asn Ser Ile Ser
690 695 700
Ala Val Asp Asn Gly Ser Leu Ala Asn Thr Pro His Leu Arg Glu Leu
705 710 715 720
His Leu Asp Asn Asn Lys Leu Thr Arg Val Pro Gly Gly Leu Ala Glu
725 730 735
His Lys Tyr Ile Gln Val Val Tyr Leu His Asn Asn Asn Ile Ser Val
740 745 750
Val Gly Ser Ser Asp Phe Cys Pro Pro Gly His Asn Thr Lys Lys Ala
755 760 765
Ser Tyr Ser Gly Val Ser Leu Phe Ser Asn Pro Val Gln Tyr Trp Glu
770 775 780
Ile Gln Pro Ser Thr Phe Arg Cys Val Tyr Val Arg Ser Ala Ile Gln
785 790 795 800
Leu Gly Asn Tyr Lys
805
<210> 30
<211> 705
<212> DNA
<213> Artificial Sequence
<220>
<223> synthetic
<400> 30
atgatgtcct ttgtctctct gctcctggtt ggcatcctat tccatgccac ccaggccgag 60
atagttttga ctcaaagccc tggaacgctc tctttgtctc ccggcgagcg ggcgaccctt 120
tcctgtaggg ctagccagag ggtatcttcc tcttacctgg catggtacca gcaaaagccc 180
ggacaagccc cccgacttct gatttatgac gcctcatccc gggcgacagg catccctgac 240
cgattttcag ggagtggctc tggtaccgat tttacgctta cgatttccag gctggagccc 300
gaggatttcg cagtgtatta ctgtcaacaa tacggcagct tgccctggac ctttggacaa 360
ggaaccaagg tagagatcaa aaggaccgtt gccgccccgt ccgtgttcat cttccctccg 420
agcgatgagc aacttaaaag tggaactgca agcgttgtat gtcttctgaa caatttctat 480
ccccgagaag ccaaggtaca gtggaaagtg gataatgccc tccaatctgg caatagccaa 540
gagtctgtca cagagcagga cagcaaggac tcaacttatt cacttagctc caccctgacg 600
ctgagtaaag cagactacga gaagcataag gtgtatgctt gtgaggttac acaccaaggc 660
ttgtcttctc ctgtcacgaa gtctttcaat aggggcgaat gctga 705
<210> 31
<211> 234
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic
<400> 31
Met Met Ser Phe Val Ser Leu Leu Leu Val Gly Ile Leu Phe His Ala
1 5 10 15
Thr Gln Ala Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu
20 25 30
Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Arg Val
35 40 45
Ser Ser Ser Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro
50 55 60
Arg Leu Leu Ile Tyr Asp Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp
65 70 75 80
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
85 90 95
Arg Leu Glu Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly
100 105 110
Ser Leu Pro Trp Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg
115 120 125
Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln
130 135 140
Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr
145 150 155 160
Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser
165 170 175
Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr
180 185 190
Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys
195 200 205
His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro
210 215 220
Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230
<210> 32
<211> 451
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic
<400> 32
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Arg Tyr
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Asn Ile Lys Gln Asp Gly Ser Glu Lys Tyr Tyr Val Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Gly Gly Trp Phe Gly Glu Leu Ala Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys
210 215 220
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Phe Glu Gly
225 230 235 240
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
245 250 255
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
260 265 270
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
275 280 285
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
290 295 300
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
305 310 315 320
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Ser Ile
325 330 335
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
340 345 350
Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser
355 360 365
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
370 375 380
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
385 390 395 400
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
405 410 415
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
420 425 430
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
435 440 445
Pro Gly Lys
450
<210> 33
<211> 215
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic
<400> 33
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Arg Val Ser Ser Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Asp Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Ser Leu Pro
85 90 95
Trp Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala
100 105 110
Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser
115 120 125
Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu
130 135 140
Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser
145 150 155 160
Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu
165 170 175
Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val
180 185 190
Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys
195 200 205
Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 34
<211> 2385
<212> DNA
<213> Artificial Sequence
<220>
<223> synthetic
<400> 34
atgatgtcct ttgtctctct gctcctggtt ggcatcctat tccatgccac ccaggcccaa 60
gtccagctcg tggaatcagg agggggtgta gtccaaccag ggcggagcct ccgacttgat 120
tgtaaagcat caggaattac attttctaat agcggaatgc attgggtccg acaagcgcca 180
ggcaagggac tggaatgggt cgcggtgata tggtatgatg gatcaaaacg ctattatgcg 240
gactctgtga agggtcgatt cactattagc agagataaca gcaagaatac tctcttcctt 300
cagatgaatt cacttagggc agaggacaca gcggtgtact attgcgcgac gaacgacgat 360
tattggggcc aagggacatt ggtaacggtg agttctgcta gtactaaagg gccttccgtc 420
ttcccactcg ccccttgttc tagaagtact agtgagtcaa cagctgcttt gggttgcttg 480
gttaaagact actttcctga acccgtgact gtgtcttgga attccggtgc tcttacttca 540
ggtgttcata cattcccagc agtattgcag agctctggct tgtattctct ctcctcagtg 600
gtgacagtac cttcctcctc tcttggcaca aaaacttaca catgtaatgt agaccataaa 660
ccatcaaaca cgaaagttga caagagagta gaaagcaagt atgggcctcc atgtcccccg 720
tgcccggcgc ccgagttcct gggtggtccg tcagtgttct tgttccctcc caagccaaaa 780
gatacattga tgataagtcg gacgccggag gtcacatgtg tagtagttga tgtctctcag 840
gaggatcctg aggtgcagtt taactggtac gtcgatggtg ttgaggtaca caacgccaaa 900
actaagccga gggaagagca gttcaattca acatatcggg tcgtgtccgt attgacagtt 960
ctgcaccaag attggttgaa cggaaaagag tataagtgca aagttagcaa taagggactt 1020
ccgtcctcaa ttgaaaaaac catttccaaa gcgaaaggcc aacctcggga acctcaggta 1080
tataccttgc cacccagcca agaagaaatg actaaaaacc aggttagttt gacatgtttg 1140
gttaaaggct tttacccgtc cgacattgcc gtcgagtggg aaagcaatgg gcagcctgaa 1200
aataactaca agacaacccc accagtattg gattccgacg gttccttctt tctttacagc 1260
cgcctcaccg tcgataagag tcggtggcaa gaggggaatg tcttttcctg tagtgtcatg 1320
cacgaagcac ttcacaacca ttacacccaa aaatcattgt ccctgtcact ggggaaatcc 1380
gggggtggcg gatccgatga ggctgcaggg ataggcccag aagttcctga tgaccgcgac 1440
ttcgagccct ccctaggccc agtgtgcccc ttccgctgtc aatgccatct tcgagtggtc 1500
cagtgttctg atttgggtct ggacaaagtg ccaaaggatc ttccccctga cacaactctg 1560
ctagacctgc aaaacaacaa aataaccgaa atcaaagatg gagactttaa gaacctgaag 1620
aaccttcacg cattgattct tgtcaacaat aaaattagca aagttagtcc tggagcattt 1680
acacctttgg tgaagttgga acgactttat ctgtccaaga atcagctgaa ggaattgcca 1740
gaaaaaatgc ccaaaactct tcaggagctg cgtgcccatg agaatgagat caccaaagtg 1800
cgaaaagtta ctttcaatgg actgaaccag atgattgtca tagaactggg caccaatccg 1860
ctgaagagct caggaattga aaatggggct ttccagggaa tgaagaagct ctcctacatc 1920
cgcattgctg ataccaatat caccagcatt cctcaaggtc ttcctccttc ccttacggaa 1980
ttacatcttg atggcaacaa aatcagcaga gttgatgcag ctagcctgaa aggactgaat 2040
aatttggcta agttgggatt gagtttcaac agcatctctg ctgttgacaa tggctctctg 2100
gccaacacgc ctcatctgag ggagcttcac ttggacaaca acaagcttac cagagtacct 2160
ggtgggctgg cagagcataa gtacatccag gttgtctacc ttcataacaa caatatctct 2220
gtagttggat caagtgactt ctgcccacct ggacacaaca ccaaaaaggc ttcttattcg 2280
ggtgtgagtc ttttcagcaa cccggtccag tactgggaga tacagccatc caccttcaga 2340
tgtgtctacg tgcgctctgc cattcaactc ggaaactata agtga 2385
<210> 35
<211> 794
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic
<400> 35
Met Met Ser Phe Val Ser Leu Leu Leu Val Gly Ile Leu Phe His Ala
1 5 10 15
Thr Gln Ala Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln
20 25 30
Pro Gly Arg Ser Leu Arg Leu Asp Cys Lys Ala Ser Gly Ile Thr Phe
35 40 45
Ser Asn Ser Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
Glu Trp Val Ala Val Ile Trp Tyr Asp Gly Ser Lys Arg Tyr Tyr Ala
65 70 75 80
Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn
85 90 95
Thr Leu Phe Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Ala Thr Asn Asp Asp Tyr Trp Gly Gln Gly Thr Leu Val
115 120 125
Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala
130 135 140
Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu
145 150 155 160
Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly
165 170 175
Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser
180 185 190
Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu
195 200 205
Gly Thr Lys Thr Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr
210 215 220
Lys Val Asp Lys Arg Val Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro
225 230 235 240
Cys Pro Ala Pro Glu Phe Leu Gly Gly Pro Ser Val Phe Leu Phe Pro
245 250 255
Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr
260 265 270
Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn
275 280 285
Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg
290 295 300
Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val
305 310 315 320
Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser
325 330 335
Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys
340 345 350
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu
355 360 365
Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
370 375 380
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
385 390 395 400
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
405 410 415
Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly
420 425 430
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
435 440 445
Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys Ser Gly Gly Gly Gly
450 455 460
Ser Asp Glu Ala Ala Gly Ile Gly Pro Glu Val Pro Asp Asp Arg Asp
465 470 475 480
Phe Glu Pro Ser Leu Gly Pro Val Cys Pro Phe Arg Cys Gln Cys His
485 490 495
Leu Arg Val Val Gln Cys Ser Asp Leu Gly Leu Asp Lys Val Pro Lys
500 505 510
Asp Leu Pro Pro Asp Thr Thr Leu Leu Asp Leu Gln Asn Asn Lys Ile
515 520 525
Thr Glu Ile Lys Asp Gly Asp Phe Lys Asn Leu Lys Asn Leu His Ala
530 535 540
Leu Ile Leu Val Asn Asn Lys Ile Ser Lys Val Ser Pro Gly Ala Phe
545 550 555 560
Thr Pro Leu Val Lys Leu Glu Arg Leu Tyr Leu Ser Lys Asn Gln Leu
565 570 575
Lys Glu Leu Pro Glu Lys Met Pro Lys Thr Leu Gln Glu Leu Arg Ala
580 585 590
His Glu Asn Glu Ile Thr Lys Val Arg Lys Val Thr Phe Asn Gly Leu
595 600 605
Asn Gln Met Ile Val Ile Glu Leu Gly Thr Asn Pro Leu Lys Ser Ser
610 615 620
Gly Ile Glu Asn Gly Ala Phe Gln Gly Met Lys Lys Leu Ser Tyr Ile
625 630 635 640
Arg Ile Ala Asp Thr Asn Ile Thr Ser Ile Pro Gln Gly Leu Pro Pro
645 650 655
Ser Leu Thr Glu Leu His Leu Asp Gly Asn Lys Ile Ser Arg Val Asp
660 665 670
Ala Ala Ser Leu Lys Gly Leu Asn Asn Leu Ala Lys Leu Gly Leu Ser
675 680 685
Phe Asn Ser Ile Ser Ala Val Asp Asn Gly Ser Leu Ala Asn Thr Pro
690 695 700
His Leu Arg Glu Leu His Leu Asp Asn Asn Lys Leu Thr Arg Val Pro
705 710 715 720
Gly Gly Leu Ala Glu His Lys Tyr Ile Gln Val Val Tyr Leu His Asn
725 730 735
Asn Asn Ile Ser Val Val Gly Ser Ser Asp Phe Cys Pro Pro Gly His
740 745 750
Asn Thr Lys Lys Ala Ser Tyr Ser Gly Val Ser Leu Phe Ser Asn Pro
755 760 765
Val Gln Tyr Trp Glu Ile Gln Pro Ser Thr Phe Arg Cys Val Tyr Val
770 775 780
Arg Ser Ala Ile Gln Leu Gly Asn Tyr Lys
785 790
<210> 36
<211> 702
<212> DNA
<213> Artificial Sequence
<220>
<223> synthetic
<400> 36
atgatgtcct ttgtctctct gctcctggtt ggcatcctat tccatgccac ccaggccgaa 60
atcgtattga ctcagtcccc tgctacactt tcactgagtc cgggtgagcg ggcgactttg 120
tcatgtcggg catcacagag tgtaagtagt tatctggcct ggtatcaaca gaaaccggga 180
caggctcctc gcctgctgat ttatgacgca agcaatcgcg cgaccggcat cccggcgagg 240
ttctcagggt ctggatcagg tactgacttt acccttacga tctcttctct cgaacctgag 300
gatttcgctg tctattactg ccagcagtct tctaactggc cgagaacatt tggtcaaggg 360
acaaaagtcg agattaagcg aactgtcgca gcgccatctg tctttatctt ccctccaagc 420
gacgaacagc ttaagagtgg caccgccagt gttgtctgcc ttctgaataa cttctatcca 480
agggaagcga aagttcagtg gaaggtggat aacgctctgc agtctgggaa ctctcaggaa 540
agtgtaacag aacaagactc caaagactca acctactctc ttagttccac gttgaccctc 600
tccaaagcgg actatgagaa gcataaggtc tacgcttgcg aggtaacaca ccaggggctg 660
agtagtccgg ttacgaagag cttcaacaga ggggaatgct ga 702
<210> 37
<211> 233
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic
<400> 37
Met Met Ser Phe Val Ser Leu Leu Leu Val Gly Ile Leu Phe His Ala
1 5 10 15
Thr Gln Ala Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu
20 25 30
Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val
35 40 45
Ser Ser Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg
50 55 60
Leu Leu Ile Tyr Asp Ala Ser Asn Arg Ala Thr Gly Ile Pro Ala Arg
65 70 75 80
Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser
85 90 95
Leu Glu Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Ser Ser Asn
100 105 110
Trp Pro Arg Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr
115 120 125
Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu
130 135 140
Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro
145 150 155 160
Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly
165 170 175
Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr
180 185 190
Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His
195 200 205
Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val
210 215 220
Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230
<210> 38
<211> 440
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic
<400> 38
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Asp Cys Lys Ala Ser Gly Ile Thr Phe Ser Asn Ser
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Trp Tyr Asp Gly Ser Lys Arg Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Phe
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Asn Asp Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser
100 105 110
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser
115 120 125
Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
130 135 140
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
145 150 155 160
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
165 170 175
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Lys
180 185 190
Thr Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr Lys Val Asp
195 200 205
Lys Arg Val Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro Ala
210 215 220
Pro Glu Phe Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro
225 230 235 240
Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val
245 250 255
Val Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val
260 265 270
Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln
275 280 285
Phe Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln
290 295 300
Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly
305 310 315 320
Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro
325 330 335
Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr
340 345 350
Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser
355 360 365
Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr
370 375 380
Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr
385 390 395 400
Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe
405 410 415
Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys
420 425 430
Ser Leu Ser Leu Ser Leu Gly Lys
435 440
<210> 39
<211> 214
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic
<400> 39
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Arg Ala Thr Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Ser Ser Asn Trp Pro Arg
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 40
<211> 2406
<212> DNA
<213> Artificial Sequence
<220>
<223> synthetic
<400> 40
atgatgtcct ttgtctctct gctcctggtt ggcatcctat tccatgccac ccaggcccag 60
gtgcaattgg tccaaagtgg ggtcgaggtc aagaagccag gagcttctgt aaaagtttca 120
tgtaaggcat ctgggtatac cttcacgaac tactatatgt attgggtgcg ccaagcgcca 180
gggcagggcc tcgaatggat gggtggcatc aatccgagca acgggggcac caactttaac 240
gaaaagttta aaaaccgggt caccttgaca acggacagta gcacgactac cgcttatatg 300
gagctgaaga gtttgcagtt tgatgatact gcggtttatt attgtgcacg cagagattat 360
aggttcgaca tgggcttcga ctactggggt caaggtacta cggtaactgt atcatctgct 420
agtacaaagg gcccttccgt tttccccctc gccccgtgca gccgctcaac gtccgaaagc 480
accgctgcac ttgggtgcct tgtaaaagac tattttccag agccagttac cgtgtcttgg 540
aatagtggcg cacttacgtc cggggtgcac acttttccgg ctgtcttgca atcctctgga 600
ctctattcct tgagtagcgt cgtaacagta ccaagtagta gtctcggcac caaaacgtat 660
acgtgcaatg ttgatcataa gcctagcaac acgaaagttg acaaaagagt tgagagtaaa 720
tatggacccc cctgtccgcc atgcccggcc cctgaattcc ttgggggccc gtctgtattt 780
cttttcccgc ccaagccgaa ggatacactg atgataagca gaacgcctga ggttacctgc 840
gtcgtggtcg acgtaagcca ggaagatcct gaggtgcaat ttaattggta tgtggacggg 900
gtcgaggttc ataatgcaaa aacaaaaccc cgagaggagc aatttaattc aacgtacaga 960
gtcgttagcg tacttacagt gctgcaccag gattggctca acgggaagga gtataagtgc 1020
aaggtgtcta ataaaggttt gccctccagt atagaaaaaa ccatctcaaa ggcgaaagga 1080
cagcctagag aacctcaagt atataccctc ccaccctccc aagaagagat gacaaagaac 1140
caagtgagtc tcacatgcct cgtcaagggt ttctacccaa gcgatatagc cgtagagtgg 1200
gaatcaaatg gtcagccgga gaataactac aaaactactc cgccagtctt ggatagcgac 1260
gggtcttttt tcctgtactc taggctgacg gtggacaagt caagatggca ggaaggaaat 1320
gtttttagct gctccgttat gcatgaggct ctccacaatc attatacaca aaaaagtttg 1380
tctctgtcat tggggaaatc cgggggtggc ggatccgatg aggctgcagg gataggccca 1440
gaagttcctg atgaccgcga cttcgagccc tccctaggcc cagtgtgccc cttccgctgt 1500
caatgccatc ttcgagtggt ccagtgttct gatttgggtc tggacaaagt gccaaaggat 1560
cttccccctg acacaactct gctagacctg caaaacaaca aaataaccga aatcaaagat 1620
ggagacttta agaacctgaa gaaccttcac gcattgattc ttgtcaacaa taaaattagc 1680
aaagttagtc ctggagcatt tacacctttg gtgaagttgg aacgacttta tctgtccaag 1740
aatcagctga aggaattgcc agaaaaaatg cccaaaactc ttcaggagct gcgtgcccat 1800
gagaatgaga tcaccaaagt gcgaaaagtt actttcaatg gactgaacca gatgattgtc 1860
atagaactgg gcaccaatcc gctgaagagc tcaggaattg aaaatggggc tttccaggga 1920
atgaagaagc tctcctacat ccgcattgct gataccaata tcaccagcat tcctcaaggt 1980
cttcctcctt cccttacgga attacatctt gatggcaaca aaatcagcag agttgatgca 2040
gctagcctga aaggactgaa taatttggct aagttgggat tgagtttcaa cagcatctct 2100
gctgttgaca atggctctct ggccaacacg cctcatctga gggagcttca cttggacaac 2160
aacaagctta ccagagtacc tggtgggctg gcagagcata agtacatcca ggttgtctac 2220
cttcataaca acaatatctc tgtagttgga tcaagtgact tctgcccacc tggacacaac 2280
accaaaaagg cttcttattc gggtgtgagt cttttcagca acccggtcca gtactgggag 2340
atacagccat ccaccttcag atgtgtctac gtgcgctctg ccattcaact cggaaactat 2400
aagtga 2406
<210> 41
<211> 801
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic
<400> 41
Met Met Ser Phe Val Ser Leu Leu Leu Val Gly Ile Leu Phe His Ala
1 5 10 15
Thr Gln Ala Gln Val Gln Leu Val Gln Ser Gly Val Glu Val Lys Lys
20 25 30
Pro Gly Ala Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe
35 40 45
Thr Asn Tyr Tyr Met Tyr Trp Val Arg Gln Ala Pro Gly Gln Gly Leu
50 55 60
Glu Trp Met Gly Gly Ile Asn Pro Ser Asn Gly Gly Thr Asn Phe Asn
65 70 75 80
Glu Lys Phe Lys Asn Arg Val Thr Leu Thr Thr Asp Ser Ser Thr Thr
85 90 95
Thr Ala Tyr Met Glu Leu Lys Ser Leu Gln Phe Asp Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Ala Arg Arg Asp Tyr Arg Phe Asp Met Gly Phe Asp Tyr
115 120 125
Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly
130 135 140
Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser
145 150 155 160
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
165 170 175
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
180 185 190
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
195 200 205
Thr Val Pro Ser Ser Ser Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val
210 215 220
Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Ser Lys
225 230 235 240
Tyr Gly Pro Pro Cys Pro Pro Cys Pro Ala Pro Glu Phe Leu Gly Gly
245 250 255
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
260 265 270
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu
275 280 285
Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
290 295 300
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg
305 310 315 320
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
325 330 335
Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu
340 345 350
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
355 360 365
Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu
370 375 380
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
385 390 395 400
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
405 410 415
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp
420 425 430
Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His
435 440 445
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu
450 455 460
Gly Lys Ser Gly Gly Gly Gly Ser Asp Glu Ala Ala Gly Ile Gly Pro
465 470 475 480
Glu Val Pro Asp Asp Arg Asp Phe Glu Pro Ser Leu Gly Pro Val Cys
485 490 495
Pro Phe Arg Cys Gln Cys His Leu Arg Val Val Gln Cys Ser Asp Leu
500 505 510
Gly Leu Asp Lys Val Pro Lys Asp Leu Pro Pro Asp Thr Thr Leu Leu
515 520 525
Asp Leu Gln Asn Asn Lys Ile Thr Glu Ile Lys Asp Gly Asp Phe Lys
530 535 540
Asn Leu Lys Asn Leu His Ala Leu Ile Leu Val Asn Asn Lys Ile Ser
545 550 555 560
Lys Val Ser Pro Gly Ala Phe Thr Pro Leu Val Lys Leu Glu Arg Leu
565 570 575
Tyr Leu Ser Lys Asn Gln Leu Lys Glu Leu Pro Glu Lys Met Pro Lys
580 585 590
Thr Leu Gln Glu Leu Arg Ala His Glu Asn Glu Ile Thr Lys Val Arg
595 600 605
Lys Val Thr Phe Asn Gly Leu Asn Gln Met Ile Val Ile Glu Leu Gly
610 615 620
Thr Asn Pro Leu Lys Ser Ser Gly Ile Glu Asn Gly Ala Phe Gln Gly
625 630 635 640
Met Lys Lys Leu Ser Tyr Ile Arg Ile Ala Asp Thr Asn Ile Thr Ser
645 650 655
Ile Pro Gln Gly Leu Pro Pro Ser Leu Thr Glu Leu His Leu Asp Gly
660 665 670
Asn Lys Ile Ser Arg Val Asp Ala Ala Ser Leu Lys Gly Leu Asn Asn
675 680 685
Leu Ala Lys Leu Gly Leu Ser Phe Asn Ser Ile Ser Ala Val Asp Asn
690 695 700
Gly Ser Leu Ala Asn Thr Pro His Leu Arg Glu Leu His Leu Asp Asn
705 710 715 720
Asn Lys Leu Thr Arg Val Pro Gly Gly Leu Ala Glu His Lys Tyr Ile
725 730 735
Gln Val Val Tyr Leu His Asn Asn Asn Ile Ser Val Val Gly Ser Ser
740 745 750
Asp Phe Cys Pro Pro Gly His Asn Thr Lys Lys Ala Ser Tyr Ser Gly
755 760 765
Val Ser Leu Phe Ser Asn Pro Val Gln Tyr Trp Glu Ile Gln Pro Ser
770 775 780
Thr Phe Arg Cys Val Tyr Val Arg Ser Ala Ile Gln Leu Gly Asn Tyr
785 790 795 800
Lys
<210> 42
<211> 714
<212> DNA
<213> Artificial Sequence
<220>
<223> synthetic
<400> 42
atgatgtcct ttgtctctct gctcctggtt ggcatcctat tccatgccac ccaggccgaa 60
attgttctta cgcagagtcc agcaactttg tcactctctc ccggcgaacg agcgacattg 120
tcctgtcgcg cgagtaaggg tgtctcaaca tctggatact catatctgca ttggtaccag 180
caaaaaccgg gacaagcgcc gcgattgctg atttatctcg cctcctacct tgaaagtggt 240
gtgcctgcga ggttctccgg tagtggatca ggcaccgatt tcactttgac catcagcagc 300
ctcgaaccag aagattttgc cgtctactac tgccaacata gcagggattt gccactgaca 360
ttcggcgggg gtacgaaagt tgagattaaa cggactgtag cggcaccttc tgtcttcatc 420
ttcccaccaa gcgatgagca gcttaaaagc ggtacagctt cagtggtgtg ccttttgaac 480
aacttttatc cgcgagaagc caaggtccaa tggaaggtgg ataacgcttt gcaatccggt 540
aactcacagg agtcagtaac agagcaagat agtaaagata gcacgtattc acttagcagt 600
acccttactc ttagcaaggc tgattatgaa aaacataagg tatatgcgtg cgaggtaacg 660
caccaaggac ttagctcccc agtgacgaag tcatttaacc ggggggagtg ctga 714
<210> 43
<211> 237
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic
<400> 43
Met Met Ser Phe Val Ser Leu Leu Leu Val Gly Ile Leu Phe His Ala
1 5 10 15
Thr Gln Ala Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu
20 25 30
Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Lys Gly Val
35 40 45
Ser Thr Ser Gly Tyr Ser Tyr Leu His Trp Tyr Gln Gln Lys Pro Gly
50 55 60
Gln Ala Pro Arg Leu Leu Ile Tyr Leu Ala Ser Tyr Leu Glu Ser Gly
65 70 75 80
Val Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu
85 90 95
Thr Ile Ser Ser Leu Glu Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln
100 105 110
His Ser Arg Asp Leu Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu
115 120 125
Ile Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser
130 135 140
Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn
145 150 155 160
Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala
165 170 175
Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys
180 185 190
Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp
195 200 205
Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu
210 215 220
Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230 235
<210> 44
<211> 447
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic
<400> 44
Gln Val Gln Leu Val Gln Ser Gly Val Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asn Tyr
20 25 30
Tyr Met Tyr Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Asn Pro Ser Asn Gly Gly Thr Asn Phe Asn Glu Lys Phe
50 55 60
Lys Asn Arg Val Thr Leu Thr Thr Asp Ser Ser Thr Thr Thr Ala Tyr
65 70 75 80
Met Glu Leu Lys Ser Leu Gln Phe Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Arg Asp Tyr Arg Phe Asp Met Gly Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val Asp His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Ser Lys Tyr Gly Pro
210 215 220
Pro Cys Pro Pro Cys Pro Ala Pro Glu Phe Leu Gly Gly Pro Ser Val
225 230 235 240
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
245 250 255
Pro Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu
260 265 270
Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
275 280 285
Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser
290 295 300
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
305 310 315 320
Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile
325 330 335
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
340 345 350
Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
355 360 365
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
370 375 380
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
385 390 395 400
Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg
405 410 415
Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
420 425 430
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys
435 440 445
<210> 45
<211> 218
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic
<400> 45
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Lys Gly Val Ser Thr Ser
20 25 30
Gly Tyr Ser Tyr Leu His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro
35 40 45
Arg Leu Leu Ile Tyr Leu Ala Ser Tyr Leu Glu Ser Gly Val Pro Ala
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
65 70 75 80
Ser Leu Glu Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln His Ser Arg
85 90 95
Asp Leu Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg
100 105 110
Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln
115 120 125
Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr
130 135 140
Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser
145 150 155 160
Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr
165 170 175
Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys
180 185 190
His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro
195 200 205
Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 46
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic
<400> 46
Gly Pro Phe Gln Gln Arg Gly Leu Phe Asp Phe Met Leu Glu
1 5 10
<210> 47
<211> 42
<212> DNA
<213> Artificial Sequence
<220>
<223> synthetic
<400> 47
ggcccgtttc aacagagagg cttatttgac tttatgctag aa 42
<210> 48
<211> 990
<212> DNA
<213> Artificial Sequence
<220>
<223> synthetic
<400> 48
gatgaggctg cagggatagg cccagaagtt cctgatgacc gcgacttcga gccctcccta 60
ggcccagtgt gccccttccg ctgtcaatgc catcttcgag tggtccagtg ttctgatttg 120
ggtctggaca aagtgccaaa ggatcttccc cctgacacaa ctctgctaga cctgcaaaac 180
aacaaaataa ccgaaatcaa agatggagac tttaagaacc tgaagaacct tcacgcattg 240
attcttgtca acaataaaat tagcaaagtt agtcctggag catttacacc tttggtgaag 300
ttggaacgac tttatctgtc caagaatcag ctgaaggaat tgccagaaaa aatgcccaaa 360
actcttcagg agctgcgtgc ccatgagaat gagatcacca aagtgcgaaa agttactttc 420
aatggactga accagatgat tgtcatagaa ctgggcacca atccgctgaa gagctcagga 480
attgaaaatg gggctttcca gggaatgaag aagctctcct acatccgcat tgctgatacc 540
aatatcacca gcattcctca aggtcttcct ccttccctta cggaattaca tcttgatggc 600
aacaaaatca gcagagttga tgcagctagc ctgaaaggac tgaataattt ggctaagttg 660
ggattgagtt tcaacagcat ctctgctgtt gacaatggct ctctggccaa cacgcctcat 720
ctgagggagc ttcacttgga caacaacaag cttaccagag tacctggtgg gctggcagag 780
cataagtaca tccaggttgt ctaccttcat aacaacaata tctctgtagt tggatcaagt 840
gacttctgcc cacctggaca caacaccaaa aaggcttctt attcgggtgt gagtcttttc 900
agcaacccgg tccagtactg ggagatacag ccatccacct tcagatgtgt ctacgtgcgc 960
tctgccattc aactcggaaa ctataagtga 990
<210> 49
<211> 2415
<212> DNA
<213> Artificial Sequence
<220>
<223> synthetic
<400> 49
atgatgtcct ttgtctctct gctcctggtt ggcatcctat tccatgccac ccaggccgag 60
gtacagcttt tggagtcagg cggggggctc gtccaacctg gggggtcact ccggttgtca 120
tgtgctgcca gtggcttcac attctcatct tacattatga tgtgggttcg acaggcccct 180
gggaaggggc tggagtgggt ttcctccatc tacccctccg ggggaattac cttctatgcc 240
gacacggtaa agggtcgctt cactataagt cgagataaca gcaaaaatac gctgtatctc 300
cagatgaact ctctcagggc tgaggacaca gctgtatatt actgcgcgcg gattaagttg 360
gggaccgtca caacagtgga ttactggggt caaggcactc tggtaaccgt atcctcagca 420
tccaccaagg gcccaagtgt attcccgctg gccccttcaa gtaaatccac gtctggcggc 480
acagccgctc tcggttgcct ggttaaggac tacttcccag aacctgtcac tgtcagttgg 540
aactcaggcg cattgacatc tggtgtccat acattccccg cagtcctgca aagctctgga 600
ctttacagtc ttagtagcgt agtgacagtc ccatcttcaa gtcttgggac ccaaacttat 660
atttgcaacg taaatcataa accctccaac actaaagtag acaaaaaagt agagccgaaa 720
tcttgcgaca aaacgcatac atgcccacca tgtcccgctc cggaactcct gggcggcccg 780
tccgtttttc tctttccccc aaagcccaag gatacgctta tgatcagcag aacaccggaa 840
gttacttgtg tagtcgttga cgtgtctcac gaagatcccg aagtcaaatt taattggtat 900
gtggatggcg tcgaagtgca caacgcaaaa accaaaccca gagaggaaca gtataacagc 960
acgtatcgag tggtctccgt acttacggtc ctccaccagg actggttgaa tggcaaggag 1020
tacaagtgca aagtgagcaa taaagcgttg ccagccccga tcgaaaaaac catcagcaag 1080
gccaagggac agcctagaga gccgcaggtt tacaccttgc cgccatcaag ggatgaactg 1140
actaaaaacc aggtatccct gacctgcctg gttaagggtt tttaccccag tgatatagcg 1200
gttgaatggg agtctaacgg gcagccagag aacaactaca aaacgacacc tcccgttctg 1260
gattccgatg gcagcttttt cttgtattct aaactcaccg tggataaatc ccgatggcag 1320
caaggcaacg tcttctcctg cagcgtgatg catgaagcct tgcacaacca ctatacccaa 1380
aagagtctca gcctgtcacc cgggaaatcc gggggtggcg gatccgatga ggctgcaggg 1440
ataggcccag aagttcctga tgaccgcgac ttcgagccct ccctaggccc agtgtgcccc 1500
ttccgctgtc aatgccatct tcgagtggtc cagtgttctg atttgggtct ggacaaagtg 1560
ccaaaggatc ttccccctga cacaactctg ctagacctgc aaaacaacaa aataaccgaa 1620
atcaaagatg gagactttaa gaacctgaag aaccttcacg cattgattct tgtcaacaat 1680
aaaattagca aagttagtcc tggagcattt acacctttgg tgaagttgga acgactttat 1740
ctgtccaaga atcagctgaa ggaattgcca gaaaaaatgc ccaaaactct tcaggagctg 1800
cgtgcccatg agaatgagat caccaaagtg cgaaaagtta ctttcaatgg actgaaccag 1860
atgattgtca tagaactggg caccaatccg ctgaagagct caggaattga aaatggggct 1920
ttccagggaa tgaagaagct ctcctacatc cgcattgctg ataccaatat caccagcatt 1980
cctcaaggtc ttcctccttc ccttacggaa ttacatcttg atggcaacaa aatcagcaga 2040
gttgatgcag ctagcctgaa aggactgaat aatttggcta agttgggatt gagtttcaac 2100
agcatctctg ctgttgacaa tggctctctg gccaacacgc ctcatctgag ggagcttcac 2160
ttggacaaca acaagcttac cagagtacct ggtgggctgg cagagcataa gtacatccag 2220
gttgtctacc ttcataacaa caatatctct gtagttggat caagtgactt ctgcccacct 2280
ggacacaaca ccaaaaaggc ttcttattcg ggtgtgagtc ttttcagcaa cccggtccag 2340
tactgggaga tacagccatc caccttcaga tgtgtctacg tgcgctctgc cattcaactc 2400
ggaaactata agtga 2415
<210> 50
<211> 804
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic
<400> 50
Met Met Ser Phe Val Ser Leu Leu Leu Val Gly Ile Leu Phe His Ala
1 5 10 15
Thr Gln Ala Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln
20 25 30
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
35 40 45
Ser Ser Tyr Ile Met Met Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
Glu Trp Val Ser Ser Ile Tyr Pro Ser Gly Gly Ile Thr Phe Tyr Ala
65 70 75 80
Asp Thr Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn
85 90 95
Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Ala Arg Ile Lys Leu Gly Thr Val Thr Thr Val Asp Tyr
115 120 125
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly
130 135 140
Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly
145 150 155 160
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
165 170 175
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
180 185 190
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
195 200 205
Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val
210 215 220
Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys
225 230 235 240
Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu
245 250 255
Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
260 265 270
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
275 280 285
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
290 295 300
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
305 310 315 320
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
325 330 335
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala
340 345 350
Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
355 360 365
Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln
370 375 380
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
385 390 395 400
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
405 410 415
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
420 425 430
Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser
435 440 445
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
450 455 460
Leu Ser Pro Gly Lys Ser Gly Gly Gly Gly Ser Asp Glu Ala Ala Gly
465 470 475 480
Ile Gly Pro Glu Val Pro Asp Asp Arg Asp Phe Glu Pro Ser Leu Gly
485 490 495
Pro Val Cys Pro Phe Arg Cys Gln Cys His Leu Arg Val Val Gln Cys
500 505 510
Ser Asp Leu Gly Leu Asp Lys Val Pro Lys Asp Leu Pro Pro Asp Thr
515 520 525
Thr Leu Leu Asp Leu Gln Asn Asn Lys Ile Thr Glu Ile Lys Asp Gly
530 535 540
Asp Phe Lys Asn Leu Lys Asn Leu His Ala Leu Ile Leu Val Asn Asn
545 550 555 560
Lys Ile Ser Lys Val Ser Pro Gly Ala Phe Thr Pro Leu Val Lys Leu
565 570 575
Glu Arg Leu Tyr Leu Ser Lys Asn Gln Leu Lys Glu Leu Pro Glu Lys
580 585 590
Met Pro Lys Thr Leu Gln Glu Leu Arg Ala His Glu Asn Glu Ile Thr
595 600 605
Lys Val Arg Lys Val Thr Phe Asn Gly Leu Asn Gln Met Ile Val Ile
610 615 620
Glu Leu Gly Thr Asn Pro Leu Lys Ser Ser Gly Ile Glu Asn Gly Ala
625 630 635 640
Phe Gln Gly Met Lys Lys Leu Ser Tyr Ile Arg Ile Ala Asp Thr Asn
645 650 655
Ile Thr Ser Ile Pro Gln Gly Leu Pro Pro Ser Leu Thr Glu Leu His
660 665 670
Leu Asp Gly Asn Lys Ile Ser Arg Val Asp Ala Ala Ser Leu Lys Gly
675 680 685
Leu Asn Asn Leu Ala Lys Leu Gly Leu Ser Phe Asn Ser Ile Ser Ala
690 695 700
Val Asp Asn Gly Ser Leu Ala Asn Thr Pro His Leu Arg Glu Leu His
705 710 715 720
Leu Asp Asn Asn Lys Leu Thr Arg Val Pro Gly Gly Leu Ala Glu His
725 730 735
Lys Tyr Ile Gln Val Val Tyr Leu His Asn Asn Asn Ile Ser Val Val
740 745 750
Gly Ser Ser Asp Phe Cys Pro Pro Gly His Asn Thr Lys Lys Ala Ser
755 760 765
Tyr Ser Gly Val Ser Leu Phe Ser Asn Pro Val Gln Tyr Trp Glu Ile
770 775 780
Gln Pro Ser Thr Phe Arg Cys Val Tyr Val Arg Ser Ala Ile Gln Leu
785 790 795 800
Gly Asn Tyr Lys
<210> 51
<211> 708
<212> DNA
<213> Artificial Sequence
<220>
<223> synthetic
<400> 51
atgatgtcct ttgtctctct gctcctggtt ggcatcctat tccatgccac ccaggcccag 60
tctgcactta cacaaccggc gtccgtttcc ggatctccag gacagagcat tactatcagt 120
tgcacgggaa cctcctcaga cgtagggggg tataattatg tgtcttggta tcaacagcat 180
cccgggaaag cccccaaact gatgatctac gatgtcagca atagaccaag cggtgtgagt 240
aatcgattta gcgggtctaa atctggtaac acagcatccc tcactattag tggactgcaa 300
gcagaagatg aggcagacta ttattgcagt agctatacgt ctagttccac ccgcgttttt 360
ggcactggga cgaaagtcac cgttctcgga caaccaaaag caaaccccac cgtgactctg 420
tttccgccta gcagcgaaga attgcaggcc aataaggcga cactcgtatg ccttatctcc 480
gacttctacc cgggcgctgt gacagtcgcg tggaaagccg acggcagccc tgttaaagct 540
ggagtcgaga ccacgaagcc gtccaagcag agtaacaata agtatgctgc atccagttat 600
ctctctctca ctccggaaca gtggaagtcc catcggtcct atagttgcca agtgacccat 660
gagggttcca ccgtagagaa aacggtagca cctaccgaat gtagttga 708
<210> 52
<211> 235
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic
<400> 52
Met Met Ser Phe Val Ser Leu Leu Leu Val Gly Ile Leu Phe His Ala
1 5 10 15
Thr Gln Ala Gln Ser Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser
20 25 30
Pro Gly Gln Ser Ile Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val
35 40 45
Gly Gly Tyr Asn Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala
50 55 60
Pro Lys Leu Met Ile Tyr Asp Val Ser Asn Arg Pro Ser Gly Val Ser
65 70 75 80
Asn Arg Phe Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile
85 90 95
Ser Gly Leu Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr
100 105 110
Thr Ser Ser Ser Thr Arg Val Phe Gly Thr Gly Thr Lys Val Thr Val
115 120 125
Leu Gly Gln Pro Lys Ala Asn Pro Thr Val Thr Leu Phe Pro Pro Ser
130 135 140
Ser Glu Glu Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser
145 150 155 160
Asp Phe Tyr Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Gly Ser
165 170 175
Pro Val Lys Ala Gly Val Glu Thr Thr Lys Pro Ser Lys Gln Ser Asn
180 185 190
Asn Lys Tyr Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp
195 200 205
Lys Ser His Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr
210 215 220
Val Glu Lys Thr Val Ala Pro Thr Glu Cys Ser
225 230 235
<210> 53
<211> 3420
<212> DNA
<213> Artificial Sequence
<220>
<223> synthetic
<400> 53
atgatgtcct ttgtctctct gctcctggtt ggcatcctat tccatgccac ccaggccgag 60
gtacagcttt tggagtcagg cggggggctc gtccaacctg gggggtcact ccggttgtca 120
tgtgctgcca gtggcttcac attctcatct tacattatga tgtgggttcg acaggcccct 180
gggaaggggc tggagtgggt ttcctccatc tacccctccg ggggaattac cttctatgcc 240
gacacggtaa agggtcgctt cactataagt cgagataaca gcaaaaatac gctgtatctc 300
cagatgaact ctctcagggc tgaggacaca gctgtatatt actgcgcgcg gattaagttg 360
gggaccgtca caacagtgga ttactggggt caaggcactc tggtaaccgt atcctcagca 420
tccaccaagg gcccaagtgt attcccgctg gccccttcaa gtaaatccac gtctggcggc 480
acagccgctc tcggttgcct ggttaaggac tacttcccag aacctgtcac tgtcagttgg 540
aactcaggcg cattgacatc tggtgtccat acattccccg cagtcctgca aagctctgga 600
ctttacagtc ttagtagcgt agtgacagtc ccatcttcaa gtcttgggac ccaaacttat 660
atttgcaacg taaatcataa accctccaac actaaagtag acaaaaaagt agagccgaaa 720
tcttgcgaca aaacgcatac atgcccacca tgtcccgctc cggaactcct gggcggcccg 780
tccgtttttc tctttccccc aaagcccaag gatacgctta tgatcagcag aacaccggaa 840
gttacttgtg tagtcgttga cgtgtctcac gaagatcccg aagtcaaatt taattggtat 900
gtggatggcg tcgaagtgca caacgcaaaa accaaaccca gagaggaaca gtataacagc 960
acgtatcgag tggtctccgt acttacggtc ctccaccagg actggttgaa tggcaaggag 1020
tacaagtgca aagtgagcaa taaagcgttg ccagccccga tcgaaaaaac catcagcaag 1080
gccaagggac agcctagaga gccgcaggtt tacaccttgc cgccatcaag ggatgaactg 1140
actaaaaacc aggtatccct gacctgcctg gttaagggtt tttaccccag tgatatagcg 1200
gttgaatggg agtctaacgg gcagccagag aacaactaca aaacgacacc tcccgttctg 1260
gattccgatg gcagcttttt cttgtattct aaactcaccg tggataaatc ccgatggcag 1320
caaggcaacg tcttctcctg cagcgtgatg catgaagcct tgcacaacca ctatacccaa 1380
aagagtctca gcctgtcacc cgggaaatcc gggggtggcg gatccgatga ggctgcaggg 1440
ataggcccag aagttcctga tgaccgcgac ttcgagccct ccctaggccc agtgtgcccc 1500
ttccgctgtc aatgccatct tcgagtggtc cagtgttctg atttgggtct ggacaaagtg 1560
ccaaaggatc ttccccctga cacaactctg ctagacctgc aaaacaacaa aataaccgaa 1620
atcaaagatg gagactttaa gaacctgaag aaccttcacg cattgattct tgtcaacaat 1680
aaaattagca aagttagtcc tggagcattt acacctttgg tgaagttgga acgactttat 1740
ctgtccaaga atcagctgaa ggaattgcca gaaaaaatgc ccaaaactct tcaggagctg 1800
cgtgcccatg agaatgagat caccaaagtg cgaaaagtta ctttcaatgg actgaaccag 1860
atgattgtca tagaactggg caccaatccg ctgaagagct caggaattga aaatggggct 1920
ttccagggaa tgaagaagct ctcctacatc cgcattgctg ataccaatat caccagcatt 1980
cctcaaggtc ttcctccttc ccttacggaa ttacatcttg atggcaacaa aatcagcaga 2040
gttgatgcag ctagcctgaa aggactgaat aatttggcta agttgggatt gagtttcaac 2100
agcatctctg ctgttgacaa tggctctctg gccaacacgc ctcatctgag ggagcttcac 2160
ttggacaaca acaagcttac cagagtacct ggtgggctgg cagagcataa gtacatccag 2220
gttgtctacc ttcataacaa caatatctct gtagttggat caagtgactt ctgcccacct 2280
ggacacaaca ccaaaaaggc ttcttattcg ggtgtgagtc ttttcagcaa cccggtccag 2340
tactgggaga tacagccatc caccttcaga tgtgtctacg tgcgctctgc cattcaactc 2400
ggaaactata agtccggggg tggcggatcc gatgaggctg cagggatagg cccagaagtt 2460
cctgatgacc gcgacttcga gccctcccta ggcccagtgt gccccttccg ctgtcaatgc 2520
catcttcgag tggtccagtg ttctgatttg ggtctggaca aagtgccaaa ggatcttccc 2580
cctgacacaa ctctgctaga cctgcaaaac aacaaaataa ccgaaatcaa agatggagac 2640
tttaagaacc tgaagaacct tcacgcattg attcttgtca acaataaaat tagcaaagtt 2700
agtcctggag catttacacc tttggtgaag ttggaacgac tttatctgtc caagaatcag 2760
ctgaaggaat tgccagaaaa aatgcccaaa actcttcagg agctgcgtgc ccatgagaat 2820
gagatcacca aagtgcgaaa agttactttc aatggactga accagatgat tgtcatagaa 2880
ctgggcacca atccgctgaa gagctcagga attgaaaatg gggctttcca gggaatgaag 2940
aagctctcct acatccgcat tgctgatacc aatatcacca gcattcctca aggtcttcct 3000
ccttccctta cggaattaca tcttgatggc aacaaaatca gcagagttga tgcagctagc 3060
ctgaaaggac tgaataattt ggctaagttg ggattgagtt tcaacagcat ctctgctgtt 3120
gacaatggct ctctggccaa cacgcctcat ctgagggagc ttcacttgga caacaacaag 3180
cttaccagag tacctggtgg gctggcagag cataagtaca tccaggttgt ctaccttcat 3240
aacaacaata tctctgtagt tggatcaagt gacttctgcc cacctggaca caacaccaaa 3300
aaggcttctt attcgggtgt gagtcttttc agcaacccgg tccagtactg ggagatacag 3360
ccatccacct tcagatgtgt ctacgtgcgc tctgccattc aactcggaaa ctataagtga 3420
<210> 54
<211> 1139
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic
<400> 54
Met Met Ser Phe Val Ser Leu Leu Leu Val Gly Ile Leu Phe His Ala
1 5 10 15
Thr Gln Ala Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln
20 25 30
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
35 40 45
Ser Ser Tyr Ile Met Met Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
Glu Trp Val Ser Ser Ile Tyr Pro Ser Gly Gly Ile Thr Phe Tyr Ala
65 70 75 80
Asp Thr Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn
85 90 95
Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Ala Arg Ile Lys Leu Gly Thr Val Thr Thr Val Asp Tyr
115 120 125
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly
130 135 140
Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly
145 150 155 160
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
165 170 175
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
180 185 190
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
195 200 205
Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val
210 215 220
Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys
225 230 235 240
Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu
245 250 255
Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
260 265 270
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
275 280 285
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
290 295 300
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
305 310 315 320
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
325 330 335
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala
340 345 350
Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
355 360 365
Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln
370 375 380
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
385 390 395 400
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
405 410 415
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
420 425 430
Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser
435 440 445
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
450 455 460
Leu Ser Pro Gly Lys Ser Gly Gly Gly Gly Ser Asp Glu Ala Ala Gly
465 470 475 480
Ile Gly Pro Glu Val Pro Asp Asp Arg Asp Phe Glu Pro Ser Leu Gly
485 490 495
Pro Val Cys Pro Phe Arg Cys Gln Cys His Leu Arg Val Val Gln Cys
500 505 510
Ser Asp Leu Gly Leu Asp Lys Val Pro Lys Asp Leu Pro Pro Asp Thr
515 520 525
Thr Leu Leu Asp Leu Gln Asn Asn Lys Ile Thr Glu Ile Lys Asp Gly
530 535 540
Asp Phe Lys Asn Leu Lys Asn Leu His Ala Leu Ile Leu Val Asn Asn
545 550 555 560
Lys Ile Ser Lys Val Ser Pro Gly Ala Phe Thr Pro Leu Val Lys Leu
565 570 575
Glu Arg Leu Tyr Leu Ser Lys Asn Gln Leu Lys Glu Leu Pro Glu Lys
580 585 590
Met Pro Lys Thr Leu Gln Glu Leu Arg Ala His Glu Asn Glu Ile Thr
595 600 605
Lys Val Arg Lys Val Thr Phe Asn Gly Leu Asn Gln Met Ile Val Ile
610 615 620
Glu Leu Gly Thr Asn Pro Leu Lys Ser Ser Gly Ile Glu Asn Gly Ala
625 630 635 640
Phe Gln Gly Met Lys Lys Leu Ser Tyr Ile Arg Ile Ala Asp Thr Asn
645 650 655
Ile Thr Ser Ile Pro Gln Gly Leu Pro Pro Ser Leu Thr Glu Leu His
660 665 670
Leu Asp Gly Asn Lys Ile Ser Arg Val Asp Ala Ala Ser Leu Lys Gly
675 680 685
Leu Asn Asn Leu Ala Lys Leu Gly Leu Ser Phe Asn Ser Ile Ser Ala
690 695 700
Val Asp Asn Gly Ser Leu Ala Asn Thr Pro His Leu Arg Glu Leu His
705 710 715 720
Leu Asp Asn Asn Lys Leu Thr Arg Val Pro Gly Gly Leu Ala Glu His
725 730 735
Lys Tyr Ile Gln Val Val Tyr Leu His Asn Asn Asn Ile Ser Val Val
740 745 750
Gly Ser Ser Asp Phe Cys Pro Pro Gly His Asn Thr Lys Lys Ala Ser
755 760 765
Tyr Ser Gly Val Ser Leu Phe Ser Asn Pro Val Gln Tyr Trp Glu Ile
770 775 780
Gln Pro Ser Thr Phe Arg Cys Val Tyr Val Arg Ser Ala Ile Gln Leu
785 790 795 800
Gly Asn Tyr Lys Ser Gly Gly Gly Gly Ser Asp Glu Ala Ala Gly Ile
805 810 815
Gly Pro Glu Val Pro Asp Asp Arg Asp Phe Glu Pro Ser Leu Gly Pro
820 825 830
Val Cys Pro Phe Arg Cys Gln Cys His Leu Arg Val Val Gln Cys Ser
835 840 845
Asp Leu Gly Leu Asp Lys Val Pro Lys Asp Leu Pro Pro Asp Thr Thr
850 855 860
Leu Leu Asp Leu Gln Asn Asn Lys Ile Thr Glu Ile Lys Asp Gly Asp
865 870 875 880
Phe Lys Asn Leu Lys Asn Leu His Ala Leu Ile Leu Val Asn Asn Lys
885 890 895
Ile Ser Lys Val Ser Pro Gly Ala Phe Thr Pro Leu Val Lys Leu Glu
900 905 910
Arg Leu Tyr Leu Ser Lys Asn Gln Leu Lys Glu Leu Pro Glu Lys Met
915 920 925
Pro Lys Thr Leu Gln Glu Leu Arg Ala His Glu Asn Glu Ile Thr Lys
930 935 940
Val Arg Lys Val Thr Phe Asn Gly Leu Asn Gln Met Ile Val Ile Glu
945 950 955 960
Leu Gly Thr Asn Pro Leu Lys Ser Ser Gly Ile Glu Asn Gly Ala Phe
965 970 975
Gln Gly Met Lys Lys Leu Ser Tyr Ile Arg Ile Ala Asp Thr Asn Ile
980 985 990
Thr Ser Ile Pro Gln Gly Leu Pro Pro Ser Leu Thr Glu Leu His Leu
995 1000 1005
Asp Gly Asn Lys Ile Ser Arg Val Asp Ala Ala Ser Leu Lys Gly
1010 1015 1020
Leu Asn Asn Leu Ala Lys Leu Gly Leu Ser Phe Asn Ser Ile Ser
1025 1030 1035
Ala Val Asp Asn Gly Ser Leu Ala Asn Thr Pro His Leu Arg Glu
1040 1045 1050
Leu His Leu Asp Asn Asn Lys Leu Thr Arg Val Pro Gly Gly Leu
1055 1060 1065
Ala Glu His Lys Tyr Ile Gln Val Val Tyr Leu His Asn Asn Asn
1070 1075 1080
Ile Ser Val Val Gly Ser Ser Asp Phe Cys Pro Pro Gly His Asn
1085 1090 1095
Thr Lys Lys Ala Ser Tyr Ser Gly Val Ser Leu Phe Ser Asn Pro
1100 1105 1110
Val Gln Tyr Trp Glu Ile Gln Pro Ser Thr Phe Arg Cys Val Tyr
1115 1120 1125
Val Arg Ser Ala Ile Gln Leu Gly Asn Tyr Lys
1130 1135
<210> 55
<211> 2373
<212> DNA
<213> Artificial Sequence
<220>
<223> synthetic
<400> 55
atgatgtcct ttgtctctct gctcctggtt ggcatcctat tccatgccac ccaggccgag 60
gtacagcttt tggagtcagg cggggggctc gtccaacctg gggggtcact ccggttgtca 120
tgtgctgcca gtggcttcac attctcatct tacattatga tgtgggttcg acaggcccct 180
gggaaggggc tggagtgggt ttcctccatc tacccctccg ggggaattac cttctatgcc 240
gacacggtaa agggtcgctt cactataagt cgagataaca gcaaaaatac gctgtatctc 300
cagatgaact ctctcagggc tgaggacaca gctgtatatt actgcgcgcg gattaagttg 360
gggaccgtca caacagtgga ttactggggt caaggcactc tggtaaccgt atcctcagca 420
tccaccaagg gcccaagtgt attcccgctg gccccttcaa gtaaatccac gtctggcggc 480
acagccgctc tcggttgcct ggttaaggac tacttcccag aacctgtcac tgtcagttgg 540
aactcaggcg cattgacatc tggtgtccat acattccccg cagtcctgca aagctctgga 600
ctttacagtc ttagtagcgt agtgacagtc ccatcttcaa gtcttgggac ccaaacttat 660
atttgcaacg taaatcataa accctccaac actaaagtag acaaaaaagt agagccgaaa 720
tcttgcgaca aaacgcatac atgcccacca tgtcccgctc cggaactcct gggcggcccg 780
tccgtttttc tctttccccc aaagcccaag gatacgctta tgatcagcag aacaccggaa 840
gttacttgtg tagtcgttga cgtgtctcac gaagatcccg aagtcaaatt taattggtat 900
gtggatggcg tcgaagtgca caacgcaaaa accaaaccca gagaggaaca gtataacagc 960
acgtatcgag tggtctccgt acttacggtc ctccaccagg actggttgaa tggcaaggag 1020
tacaagtgca aagtgagcaa taaagcgttg ccagccccga tcgaaaaaac catcagcaag 1080
gccaagggac agcctagaga gccgcaggtt tacaccttgc cgccatcaag ggatgaactg 1140
actaaaaacc aggtatccct gacctgcctg gttaagggtt tttaccccag tgatatagcg 1200
gttgaatggg agtctaacgg gcagccagag aacaactaca aaacgacacc tcccgttctg 1260
gattccgatg gcagcttttt cttgtattct aaactcaccg tggataaatc ccgatggcag 1320
caaggcaacg tcttctcctg cagcgtgatg catgaagcct tgcacaacca ctatacccaa 1380
aagagtctca gcctgtcacc cgggaaatcc gggggtggcg gatccgactt cgagccctcc 1440
ctaggcccag tgtgcccctt ccgctgtcaa tgccatcttc gagtggtcca gtgttctgat 1500
ttgggtctgg acaaagtgcc aaaggatctt ccccctgaca caactctgct agacctgcaa 1560
aacaacaaaa taaccgaaat caaagatgga gactttaaga acctgaagaa ccttcacgca 1620
ttgattcttg tcaacaataa aattagcaaa gttagtcctg gagcatttac acctttggtg 1680
aagttggaac gactttatct gtccaagaat cagctgaagg aattgccaga aaaaatgccc 1740
aaaactcttc aggagctgcg tgcccatgag aatgagatca ccaaagtgcg aaaagttact 1800
ttcaatggac tgaaccagat gattgtcata gaactgggca ccaatccgct gaagagctca 1860
ggaattgaaa atggggcttt ccagggaatg aagaagctct cctacatccg cattgctgat 1920
accaatatca ccagcattcc tcaaggtctt cctccttccc ttacggaatt acatcttgat 1980
ggcaacaaaa tcagcagagt tgatgcagct agcctgaaag gactgaataa tttggctaag 2040
ttgggattga gtttcaacag catctctgct gttgacaatg gctctctggc caacacgcct 2100
catctgaggg agcttcactt ggacaacaac aagcttacca gagtacctgg tgggctggca 2160
gagcataagt acatccaggt tgtctacctt cataacaaca atatctctgt agttggatca 2220
agtgacttct gcccacctgg acacaacacc aaaaaggctt cttattcggg tgtgagtctt 2280
ttcagcaacc cggtccagta ctgggagata cagccatcca ccttcagatg tgtctacgtg 2340
cgctctgcca ttcaactcgg aaactataag tga 2373
<210> 56
<211> 790
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic
<400> 56
Met Met Ser Phe Val Ser Leu Leu Leu Val Gly Ile Leu Phe His Ala
1 5 10 15
Thr Gln Ala Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln
20 25 30
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
35 40 45
Ser Ser Tyr Ile Met Met Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
Glu Trp Val Ser Ser Ile Tyr Pro Ser Gly Gly Ile Thr Phe Tyr Ala
65 70 75 80
Asp Thr Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn
85 90 95
Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Ala Arg Ile Lys Leu Gly Thr Val Thr Thr Val Asp Tyr
115 120 125
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly
130 135 140
Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly
145 150 155 160
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
165 170 175
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
180 185 190
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
195 200 205
Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val
210 215 220
Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys
225 230 235 240
Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu
245 250 255
Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
260 265 270
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
275 280 285
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
290 295 300
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
305 310 315 320
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
325 330 335
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala
340 345 350
Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
355 360 365
Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln
370 375 380
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
385 390 395 400
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
405 410 415
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
420 425 430
Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser
435 440 445
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
450 455 460
Leu Ser Pro Gly Lys Ser Gly Gly Gly Gly Ser Asp Phe Glu Pro Ser
465 470 475 480
Leu Gly Pro Val Cys Pro Phe Arg Cys Gln Cys His Leu Arg Val Val
485 490 495
Gln Cys Ser Asp Leu Gly Leu Asp Lys Val Pro Lys Asp Leu Pro Pro
500 505 510
Asp Thr Thr Leu Leu Asp Leu Gln Asn Asn Lys Ile Thr Glu Ile Lys
515 520 525
Asp Gly Asp Phe Lys Asn Leu Lys Asn Leu His Ala Leu Ile Leu Val
530 535 540
Asn Asn Lys Ile Ser Lys Val Ser Pro Gly Ala Phe Thr Pro Leu Val
545 550 555 560
Lys Leu Glu Arg Leu Tyr Leu Ser Lys Asn Gln Leu Lys Glu Leu Pro
565 570 575
Glu Lys Met Pro Lys Thr Leu Gln Glu Leu Arg Ala His Glu Asn Glu
580 585 590
Ile Thr Lys Val Arg Lys Val Thr Phe Asn Gly Leu Asn Gln Met Ile
595 600 605
Val Ile Glu Leu Gly Thr Asn Pro Leu Lys Ser Ser Gly Ile Glu Asn
610 615 620
Gly Ala Phe Gln Gly Met Lys Lys Leu Ser Tyr Ile Arg Ile Ala Asp
625 630 635 640
Thr Asn Ile Thr Ser Ile Pro Gln Gly Leu Pro Pro Ser Leu Thr Glu
645 650 655
Leu His Leu Asp Gly Asn Lys Ile Ser Arg Val Asp Ala Ala Ser Leu
660 665 670
Lys Gly Leu Asn Asn Leu Ala Lys Leu Gly Leu Ser Phe Asn Ser Ile
675 680 685
Ser Ala Val Asp Asn Gly Ser Leu Ala Asn Thr Pro His Leu Arg Glu
690 695 700
Leu His Leu Asp Asn Asn Lys Leu Thr Arg Val Pro Gly Gly Leu Ala
705 710 715 720
Glu His Lys Tyr Ile Gln Val Val Tyr Leu His Asn Asn Asn Ile Ser
725 730 735
Val Val Gly Ser Ser Asp Phe Cys Pro Pro Gly His Asn Thr Lys Lys
740 745 750
Ala Ser Tyr Ser Gly Val Ser Leu Phe Ser Asn Pro Val Gln Tyr Trp
755 760 765
Glu Ile Gln Pro Ser Thr Phe Arg Cys Val Tyr Val Arg Ser Ala Ile
770 775 780
Gln Leu Gly Asn Tyr Lys
785 790
<210> 57
<211> 3336
<212> DNA
<213> Artificial Sequence
<220>
<223> synthetic
<400> 57
atgatgtcct ttgtctctct gctcctggtt ggcatcctat tccatgccac ccaggccgag 60
gtacagcttt tggagtcagg cggggggctc gtccaacctg gggggtcact ccggttgtca 120
tgtgctgcca gtggcttcac attctcatct tacattatga tgtgggttcg acaggcccct 180
gggaaggggc tggagtgggt ttcctccatc tacccctccg ggggaattac cttctatgcc 240
gacacggtaa agggtcgctt cactataagt cgagataaca gcaaaaatac gctgtatctc 300
cagatgaact ctctcagggc tgaggacaca gctgtatatt actgcgcgcg gattaagttg 360
gggaccgtca caacagtgga ttactggggt caaggcactc tggtaaccgt atcctcagca 420
tccaccaagg gcccaagtgt attcccgctg gccccttcaa gtaaatccac gtctggcggc 480
acagccgctc tcggttgcct ggttaaggac tacttcccag aacctgtcac tgtcagttgg 540
aactcaggcg cattgacatc tggtgtccat acattccccg cagtcctgca aagctctgga 600
ctttacagtc ttagtagcgt agtgacagtc ccatcttcaa gtcttgggac ccaaacttat 660
atttgcaacg taaatcataa accctccaac actaaagtag acaaaaaagt agagccgaaa 720
tcttgcgaca aaacgcatac atgcccacca tgtcccgctc cggaactcct gggcggcccg 780
tccgtttttc tctttccccc aaagcccaag gatacgctta tgatcagcag aacaccggaa 840
gttacttgtg tagtcgttga cgtgtctcac gaagatcccg aagtcaaatt taattggtat 900
gtggatggcg tcgaagtgca caacgcaaaa accaaaccca gagaggaaca gtataacagc 960
acgtatcgag tggtctccgt acttacggtc ctccaccagg actggttgaa tggcaaggag 1020
tacaagtgca aagtgagcaa taaagcgttg ccagccccga tcgaaaaaac catcagcaag 1080
gccaagggac agcctagaga gccgcaggtt tacaccttgc cgccatcaag ggatgaactg 1140
actaaaaacc aggtatccct gacctgcctg gttaagggtt tttaccccag tgatatagcg 1200
gttgaatggg agtctaacgg gcagccagag aacaactaca aaacgacacc tcccgttctg 1260
gattccgatg gcagcttttt cttgtattct aaactcaccg tggataaatc ccgatggcag 1320
caaggcaacg tcttctcctg cagcgtgatg catgaagcct tgcacaacca ctatacccaa 1380
aagagtctca gcctgtcacc cgggaaatcc gggggtggcg gatccgactt cgagccctcc 1440
ctaggcccag tgtgcccctt ccgctgtcaa tgccatcttc gagtggtcca gtgttctgat 1500
ttgggtctgg acaaagtgcc aaaggatctt ccccctgaca caactctgct agacctgcaa 1560
aacaacaaaa taaccgaaat caaagatgga gactttaaga acctgaagaa ccttcacgca 1620
ttgattcttg tcaacaataa aattagcaaa gttagtcctg gagcatttac acctttggtg 1680
aagttggaac gactttatct gtccaagaat cagctgaagg aattgccaga aaaaatgccc 1740
aaaactcttc aggagctgcg tgcccatgag aatgagatca ccaaagtgcg aaaagttact 1800
ttcaatggac tgaaccagat gattgtcata gaactgggca ccaatccgct gaagagctca 1860
ggaattgaaa atggggcttt ccagggaatg aagaagctct cctacatccg cattgctgat 1920
accaatatca ccagcattcc tcaaggtctt cctccttccc ttacggaatt acatcttgat 1980
ggcaacaaaa tcagcagagt tgatgcagct agcctgaaag gactgaataa tttggctaag 2040
ttgggattga gtttcaacag catctctgct gttgacaatg gctctctggc caacacgcct 2100
catctgaggg agcttcactt ggacaacaac aagcttacca gagtacctgg tgggctggca 2160
gagcataagt acatccaggt tgtctacctt cataacaaca atatctctgt agttggatca 2220
agtgacttct gcccacctgg acacaacacc aaaaaggctt cttattcggg tgtgagtctt 2280
ttcagcaacc cggtccagta ctgggagata cagccatcca ccttcagatg tgtctacgtg 2340
cgctctgcca ttcaactcgg aaactataag tccgggggtg gcggatccga cttcgagccc 2400
tccctaggcc cagtgtgccc cttccgctgt caatgccatc ttcgagtggt ccagtgttct 2460
gatttgggtc tggacaaagt gccaaaggat cttccccctg acacaactct gctagacctg 2520
caaaacaaca aaataaccga aatcaaagat ggagacttta agaacctgaa gaaccttcac 2580
gcattgattc ttgtcaacaa taaaattagc aaagttagtc ctggagcatt tacacctttg 2640
gtgaagttgg aacgacttta tctgtccaag aatcagctga aggaattgcc agaaaaaatg 2700
cccaaaactc ttcaggagct gcgtgcccat gagaatgaga tcaccaaagt gcgaaaagtt 2760
actttcaatg gactgaacca gatgattgtc atagaactgg gcaccaatcc gctgaagagc 2820
tcaggaattg aaaatggggc tttccaggga atgaagaagc tctcctacat ccgcattgct 2880
gataccaata tcaccagcat tcctcaaggt cttcctcctt cccttacgga attacatctt 2940
gatggcaaca aaatcagcag agttgatgca gctagcctga aaggactgaa taatttggct 3000
aagttgggat tgagtttcaa cagcatctct gctgttgaca atggctctct ggccaacacg 3060
cctcatctga gggagcttca cttggacaac aacaagctta ccagagtacc tggtgggctg 3120
gcagagcata agtacatcca ggttgtctac cttcataaca acaatatctc tgtagttgga 3180
tcaagtgact tctgcccacc tggacacaac accaaaaagg cttcttattc gggtgtgagt 3240
cttttcagca acccggtcca gtactgggag atacagccat ccaccttcag atgtgtctac 3300
gtgcgctctg ccattcaact cggaaactat aagtga 3336
<210> 58
<211> 1111
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic
<400> 58
Met Met Ser Phe Val Ser Leu Leu Leu Val Gly Ile Leu Phe His Ala
1 5 10 15
Thr Gln Ala Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln
20 25 30
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
35 40 45
Ser Ser Tyr Ile Met Met Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
Glu Trp Val Ser Ser Ile Tyr Pro Ser Gly Gly Ile Thr Phe Tyr Ala
65 70 75 80
Asp Thr Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn
85 90 95
Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Ala Arg Ile Lys Leu Gly Thr Val Thr Thr Val Asp Tyr
115 120 125
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly
130 135 140
Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly
145 150 155 160
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
165 170 175
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
180 185 190
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
195 200 205
Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val
210 215 220
Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys
225 230 235 240
Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu
245 250 255
Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
260 265 270
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
275 280 285
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
290 295 300
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
305 310 315 320
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
325 330 335
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala
340 345 350
Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
355 360 365
Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln
370 375 380
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
385 390 395 400
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
405 410 415
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
420 425 430
Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser
435 440 445
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
450 455 460
Leu Ser Pro Gly Lys Ser Gly Gly Gly Gly Ser Asp Phe Glu Pro Ser
465 470 475 480
Leu Gly Pro Val Cys Pro Phe Arg Cys Gln Cys His Leu Arg Val Val
485 490 495
Gln Cys Ser Asp Leu Gly Leu Asp Lys Val Pro Lys Asp Leu Pro Pro
500 505 510
Asp Thr Thr Leu Leu Asp Leu Gln Asn Asn Lys Ile Thr Glu Ile Lys
515 520 525
Asp Gly Asp Phe Lys Asn Leu Lys Asn Leu His Ala Leu Ile Leu Val
530 535 540
Asn Asn Lys Ile Ser Lys Val Ser Pro Gly Ala Phe Thr Pro Leu Val
545 550 555 560
Lys Leu Glu Arg Leu Tyr Leu Ser Lys Asn Gln Leu Lys Glu Leu Pro
565 570 575
Glu Lys Met Pro Lys Thr Leu Gln Glu Leu Arg Ala His Glu Asn Glu
580 585 590
Ile Thr Lys Val Arg Lys Val Thr Phe Asn Gly Leu Asn Gln Met Ile
595 600 605
Val Ile Glu Leu Gly Thr Asn Pro Leu Lys Ser Ser Gly Ile Glu Asn
610 615 620
Gly Ala Phe Gln Gly Met Lys Lys Leu Ser Tyr Ile Arg Ile Ala Asp
625 630 635 640
Thr Asn Ile Thr Ser Ile Pro Gln Gly Leu Pro Pro Ser Leu Thr Glu
645 650 655
Leu His Leu Asp Gly Asn Lys Ile Ser Arg Val Asp Ala Ala Ser Leu
660 665 670
Lys Gly Leu Asn Asn Leu Ala Lys Leu Gly Leu Ser Phe Asn Ser Ile
675 680 685
Ser Ala Val Asp Asn Gly Ser Leu Ala Asn Thr Pro His Leu Arg Glu
690 695 700
Leu His Leu Asp Asn Asn Lys Leu Thr Arg Val Pro Gly Gly Leu Ala
705 710 715 720
Glu His Lys Tyr Ile Gln Val Val Tyr Leu His Asn Asn Asn Ile Ser
725 730 735
Val Val Gly Ser Ser Asp Phe Cys Pro Pro Gly His Asn Thr Lys Lys
740 745 750
Ala Ser Tyr Ser Gly Val Ser Leu Phe Ser Asn Pro Val Gln Tyr Trp
755 760 765
Glu Ile Gln Pro Ser Thr Phe Arg Cys Val Tyr Val Arg Ser Ala Ile
770 775 780
Gln Leu Gly Asn Tyr Lys Ser Gly Gly Gly Gly Ser Asp Phe Glu Pro
785 790 795 800
Ser Leu Gly Pro Val Cys Pro Phe Arg Cys Gln Cys His Leu Arg Val
805 810 815
Val Gln Cys Ser Asp Leu Gly Leu Asp Lys Val Pro Lys Asp Leu Pro
820 825 830
Pro Asp Thr Thr Leu Leu Asp Leu Gln Asn Asn Lys Ile Thr Glu Ile
835 840 845
Lys Asp Gly Asp Phe Lys Asn Leu Lys Asn Leu His Ala Leu Ile Leu
850 855 860
Val Asn Asn Lys Ile Ser Lys Val Ser Pro Gly Ala Phe Thr Pro Leu
865 870 875 880
Val Lys Leu Glu Arg Leu Tyr Leu Ser Lys Asn Gln Leu Lys Glu Leu
885 890 895
Pro Glu Lys Met Pro Lys Thr Leu Gln Glu Leu Arg Ala His Glu Asn
900 905 910
Glu Ile Thr Lys Val Arg Lys Val Thr Phe Asn Gly Leu Asn Gln Met
915 920 925
Ile Val Ile Glu Leu Gly Thr Asn Pro Leu Lys Ser Ser Gly Ile Glu
930 935 940
Asn Gly Ala Phe Gln Gly Met Lys Lys Leu Ser Tyr Ile Arg Ile Ala
945 950 955 960
Asp Thr Asn Ile Thr Ser Ile Pro Gln Gly Leu Pro Pro Ser Leu Thr
965 970 975
Glu Leu His Leu Asp Gly Asn Lys Ile Ser Arg Val Asp Ala Ala Ser
980 985 990
Leu Lys Gly Leu Asn Asn Leu Ala Lys Leu Gly Leu Ser Phe Asn Ser
995 1000 1005
Ile Ser Ala Val Asp Asn Gly Ser Leu Ala Asn Thr Pro His Leu
1010 1015 1020
Arg Glu Leu His Leu Asp Asn Asn Lys Leu Thr Arg Val Pro Gly
1025 1030 1035
Gly Leu Ala Glu His Lys Tyr Ile Gln Val Val Tyr Leu His Asn
1040 1045 1050
Asn Asn Ile Ser Val Val Gly Ser Ser Asp Phe Cys Pro Pro Gly
1055 1060 1065
His Asn Thr Lys Lys Ala Ser Tyr Ser Gly Val Ser Leu Phe Ser
1070 1075 1080
Asn Pro Val Gln Tyr Trp Glu Ile Gln Pro Ser Thr Phe Arg Cys
1085 1090 1095
Val Tyr Val Arg Ser Ala Ile Gln Leu Gly Asn Tyr Lys
1100 1105 1110
<210> 59
<211> 1746
<212> DNA
<213> Artificial Sequence
<220>
<223> synthetic
<400> 59
atgatgtcct ttgtctctct gctcctggtt ggcatcctat tccatgccac ccaggccgag 60
gtacagcttt tggagtcagg cggggggctc gtccaacctg gggggtcact ccggttgtca 120
tgtgctgcca gtggcttcac attctcatct tacattatga tgtgggttcg acaggcccct 180
gggaaggggc tggagtgggt ttcctccatc tacccctccg ggggaattac cttctatgcc 240
gacacggtaa agggtcgctt cactataagt cgagataaca gcaaaaatac gctgtatctc 300
cagatgaact ctctcagggc tgaggacaca gctgtatatt actgcgcgcg gattaagttg 360
gggaccgtca caacagtgga ttactggggt caaggcactc tggtaaccgt atcctcagca 420
tccaccaagg gcccaagtgt attcccgctg gccccttcaa gtaaatccac gtctggcggc 480
acagccgctc tcggttgcct ggttaaggac tacttcccag aacctgtcac tgtcagttgg 540
aactcaggcg cattgacatc tggtgtccat acattccccg cagtcctgca aagctctgga 600
ctttacagtc ttagtagcgt agtgacagtc ccatcttcaa gtcttgggac ccaaacttat 660
atttgcaacg taaatcataa accctccaac actaaagtag acaaaaaagt agagccgaaa 720
tcttgcgaca aaacgcatac atgcccacca tgtcccgctc cggaactcct gggcggcccg 780
tccgtttttc tctttccccc aaagcccaag gatacgctta tgatcagcag aacaccggaa 840
gttacttgtg tagtcgttga cgtgtctcac gaagatcccg aagtcaaatt taattggtat 900
gtggatggcg tcgaagtgca caacgcaaaa accaaaccca gagaggaaca gtataacagc 960
acgtatcgag tggtctccgt acttacggtc ctccaccagg actggttgaa tggcaaggag 1020
tacaagtgca aagtgagcaa taaagcgttg ccagccccga tcgaaaaaac catcagcaag 1080
gccaagggac agcctagaga gccgcaggtt tacaccttgc cgccatcaag ggatgaactg 1140
actaaaaacc aggtatccct gacctgcctg gttaagggtt tttaccccag tgatatagcg 1200
gttgaatggg agtctaacgg gcagccagag aacaactaca aaacgacacc tcccgttctg 1260
gattccgatg gcagcttttt cttgtattct aaactcaccg tggataaatc ccgatggcag 1320
caaggcaacg tcttctcctg cagcgtgatg catgaagcct tgcacaacca ctatacccaa 1380
aagagtctca gcctgtcacc cgggaaatcc gggggtggcg gatccctgcg tgcccatgag 1440
aatgagatca ccaaagtgcg aaaagttact ttcaatggac tgaaccagat gattgtcata 1500
gaactgggca ccaatccgct gaagagctca ggaattgaaa atggggcttt ccagggaatg 1560
aagaagctct cctacatccg cattgctgat accaatatca ccagcattcc tcaaggtctt 1620
cctccttccc ttacggaatt acatcttgat ggcaacaaaa tcagcagagt tgatgcagct 1680
agcctgaaag gactgaataa tttggctaag ttgggattga gtttcaacag catctctgct 1740
gtttga 1746
<210> 60
<211> 581
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic
<400> 60
Met Met Ser Phe Val Ser Leu Leu Leu Val Gly Ile Leu Phe His Ala
1 5 10 15
Thr Gln Ala Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln
20 25 30
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
35 40 45
Ser Ser Tyr Ile Met Met Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
Glu Trp Val Ser Ser Ile Tyr Pro Ser Gly Gly Ile Thr Phe Tyr Ala
65 70 75 80
Asp Thr Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn
85 90 95
Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Ala Arg Ile Lys Leu Gly Thr Val Thr Thr Val Asp Tyr
115 120 125
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly
130 135 140
Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly
145 150 155 160
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
165 170 175
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
180 185 190
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
195 200 205
Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val
210 215 220
Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys
225 230 235 240
Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu
245 250 255
Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
260 265 270
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
275 280 285
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
290 295 300
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
305 310 315 320
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
325 330 335
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala
340 345 350
Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
355 360 365
Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln
370 375 380
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
385 390 395 400
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
405 410 415
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
420 425 430
Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser
435 440 445
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
450 455 460
Leu Ser Pro Gly Lys Ser Gly Gly Gly Gly Ser Leu Arg Ala His Glu
465 470 475 480
Asn Glu Ile Thr Lys Val Arg Lys Val Thr Phe Asn Gly Leu Asn Gln
485 490 495
Met Ile Val Ile Glu Leu Gly Thr Asn Pro Leu Lys Ser Ser Gly Ile
500 505 510
Glu Asn Gly Ala Phe Gln Gly Met Lys Lys Leu Ser Tyr Ile Arg Ile
515 520 525
Ala Asp Thr Asn Ile Thr Ser Ile Pro Gln Gly Leu Pro Pro Ser Leu
530 535 540
Thr Glu Leu His Leu Asp Gly Asn Lys Ile Ser Arg Val Asp Ala Ala
545 550 555 560
Ser Leu Lys Gly Leu Asn Asn Leu Ala Lys Leu Gly Leu Ser Phe Asn
565 570 575
Ser Ile Ser Ala Val
580
<210> 61
<211> 2082
<212> DNA
<213> Artificial Sequence
<220>
<223> synthetic
<400> 61
atgatgtcct ttgtctctct gctcctggtt ggcatcctat tccatgccac ccaggccgag 60
gtacagcttt tggagtcagg cggggggctc gtccaacctg gggggtcact ccggttgtca 120
tgtgctgcca gtggcttcac attctcatct tacattatga tgtgggttcg acaggcccct 180
gggaaggggc tggagtgggt ttcctccatc tacccctccg ggggaattac cttctatgcc 240
gacacggtaa agggtcgctt cactataagt cgagataaca gcaaaaatac gctgtatctc 300
cagatgaact ctctcagggc tgaggacaca gctgtatatt actgcgcgcg gattaagttg 360
gggaccgtca caacagtgga ttactggggt caaggcactc tggtaaccgt atcctcagca 420
tccaccaagg gcccaagtgt attcccgctg gccccttcaa gtaaatccac gtctggcggc 480
acagccgctc tcggttgcct ggttaaggac tacttcccag aacctgtcac tgtcagttgg 540
aactcaggcg cattgacatc tggtgtccat acattccccg cagtcctgca aagctctgga 600
ctttacagtc ttagtagcgt agtgacagtc ccatcttcaa gtcttgggac ccaaacttat 660
atttgcaacg taaatcataa accctccaac actaaagtag acaaaaaagt agagccgaaa 720
tcttgcgaca aaacgcatac atgcccacca tgtcccgctc cggaactcct gggcggcccg 780
tccgtttttc tctttccccc aaagcccaag gatacgctta tgatcagcag aacaccggaa 840
gttacttgtg tagtcgttga cgtgtctcac gaagatcccg aagtcaaatt taattggtat 900
gtggatggcg tcgaagtgca caacgcaaaa accaaaccca gagaggaaca gtataacagc 960
acgtatcgag tggtctccgt acttacggtc ctccaccagg actggttgaa tggcaaggag 1020
tacaagtgca aagtgagcaa taaagcgttg ccagccccga tcgaaaaaac catcagcaag 1080
gccaagggac agcctagaga gccgcaggtt tacaccttgc cgccatcaag ggatgaactg 1140
actaaaaacc aggtatccct gacctgcctg gttaagggtt tttaccccag tgatatagcg 1200
gttgaatggg agtctaacgg gcagccagag aacaactaca aaacgacacc tcccgttctg 1260
gattccgatg gcagcttttt cttgtattct aaactcaccg tggataaatc ccgatggcag 1320
caaggcaacg tcttctcctg cagcgtgatg catgaagcct tgcacaacca ctatacccaa 1380
aagagtctca gcctgtcacc cgggaaatcc gggggtggcg gatccctgcg tgcccatgag 1440
aatgagatca ccaaagtgcg aaaagttact ttcaatggac tgaaccagat gattgtcata 1500
gaactgggca ccaatccgct gaagagctca ggaattgaaa atggggcttt ccagggaatg 1560
aagaagctct cctacatccg cattgctgat accaatatca ccagcattcc tcaaggtctt 1620
cctccttccc ttacggaatt acatcttgat ggcaacaaaa tcagcagagt tgatgcagct 1680
agcctgaaag gactgaataa tttggctaag ttgggattga gtttcaacag catctctgct 1740
gtttccgggg gtggcggatc cctgcgtgcc catgagaatg agatcaccaa agtgcgaaaa 1800
gttactttca atggactgaa ccagatgatt gtcatagaac tgggcaccaa tccgctgaag 1860
agctcaggaa ttgaaaatgg ggctttccag ggaatgaaga agctctccta catccgcatt 1920
gctgatacca atatcaccag cattcctcaa ggtcttcctc cttcccttac ggaattacat 1980
cttgatggca acaaaatcag cagagttgat gcagctagcc tgaaaggact gaataatttg 2040
gctaagttgg gattgagttt caacagcatc tctgctgttt ga 2082
<210> 62
<211> 693
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic
<400> 62
Met Met Ser Phe Val Ser Leu Leu Leu Val Gly Ile Leu Phe His Ala
1 5 10 15
Thr Gln Ala Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln
20 25 30
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
35 40 45
Ser Ser Tyr Ile Met Met Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
Glu Trp Val Ser Ser Ile Tyr Pro Ser Gly Gly Ile Thr Phe Tyr Ala
65 70 75 80
Asp Thr Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn
85 90 95
Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Ala Arg Ile Lys Leu Gly Thr Val Thr Thr Val Asp Tyr
115 120 125
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly
130 135 140
Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly
145 150 155 160
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
165 170 175
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
180 185 190
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
195 200 205
Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val
210 215 220
Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys
225 230 235 240
Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu
245 250 255
Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
260 265 270
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
275 280 285
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
290 295 300
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
305 310 315 320
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
325 330 335
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala
340 345 350
Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
355 360 365
Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln
370 375 380
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
385 390 395 400
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
405 410 415
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
420 425 430
Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser
435 440 445
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
450 455 460
Leu Ser Pro Gly Lys Ser Gly Gly Gly Gly Ser Leu Arg Ala His Glu
465 470 475 480
Asn Glu Ile Thr Lys Val Arg Lys Val Thr Phe Asn Gly Leu Asn Gln
485 490 495
Met Ile Val Ile Glu Leu Gly Thr Asn Pro Leu Lys Ser Ser Gly Ile
500 505 510
Glu Asn Gly Ala Phe Gln Gly Met Lys Lys Leu Ser Tyr Ile Arg Ile
515 520 525
Ala Asp Thr Asn Ile Thr Ser Ile Pro Gln Gly Leu Pro Pro Ser Leu
530 535 540
Thr Glu Leu His Leu Asp Gly Asn Lys Ile Ser Arg Val Asp Ala Ala
545 550 555 560
Ser Leu Lys Gly Leu Asn Asn Leu Ala Lys Leu Gly Leu Ser Phe Asn
565 570 575
Ser Ile Ser Ala Val Ser Gly Gly Gly Gly Ser Leu Arg Ala His Glu
580 585 590
Asn Glu Ile Thr Lys Val Arg Lys Val Thr Phe Asn Gly Leu Asn Gln
595 600 605
Met Ile Val Ile Glu Leu Gly Thr Asn Pro Leu Lys Ser Ser Gly Ile
610 615 620
Glu Asn Gly Ala Phe Gln Gly Met Lys Lys Leu Ser Tyr Ile Arg Ile
625 630 635 640
Ala Asp Thr Asn Ile Thr Ser Ile Pro Gln Gly Leu Pro Pro Ser Leu
645 650 655
Thr Glu Leu His Leu Asp Gly Asn Lys Ile Ser Arg Val Asp Ala Ala
660 665 670
Ser Leu Lys Gly Leu Asn Asn Leu Ala Lys Leu Gly Leu Ser Phe Asn
675 680 685
Ser Ile Ser Ala Val
690
<210> 63
<211> 315
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic
<400> 63
Asp Phe Glu Pro Ser Leu Gly Pro Val Cys Pro Phe Arg Cys Gln Cys
1 5 10 15
His Leu Arg Val Val Gln Cys Ser Asp Leu Gly Leu Asp Lys Val Pro
20 25 30
Lys Asp Leu Pro Pro Asp Thr Thr Leu Leu Asp Leu Gln Asn Asn Lys
35 40 45
Ile Thr Glu Ile Lys Asp Gly Asp Phe Lys Asn Leu Lys Asn Leu His
50 55 60
Ala Leu Ile Leu Val Asn Asn Lys Ile Ser Lys Val Ser Pro Gly Ala
65 70 75 80
Phe Thr Pro Leu Val Lys Leu Glu Arg Leu Tyr Leu Ser Lys Asn Gln
85 90 95
Leu Lys Glu Leu Pro Glu Lys Met Pro Lys Thr Leu Gln Glu Leu Arg
100 105 110
Ala His Glu Asn Glu Ile Thr Lys Val Arg Lys Val Thr Phe Asn Gly
115 120 125
Leu Asn Gln Met Ile Val Ile Glu Leu Gly Thr Asn Pro Leu Lys Ser
130 135 140
Ser Gly Ile Glu Asn Gly Ala Phe Gln Gly Met Lys Lys Leu Ser Tyr
145 150 155 160
Ile Arg Ile Ala Asp Thr Asn Ile Thr Ser Ile Pro Gln Gly Leu Pro
165 170 175
Pro Ser Leu Thr Glu Leu His Leu Asp Gly Asn Lys Ile Ser Arg Val
180 185 190
Asp Ala Ala Ser Leu Lys Gly Leu Asn Asn Leu Ala Lys Leu Gly Leu
195 200 205
Ser Phe Asn Ser Ile Ser Ala Val Asp Asn Gly Ser Leu Ala Asn Thr
210 215 220
Pro His Leu Arg Glu Leu His Leu Asp Asn Asn Lys Leu Thr Arg Val
225 230 235 240
Pro Gly Gly Leu Ala Glu His Lys Tyr Ile Gln Val Val Tyr Leu His
245 250 255
Asn Asn Asn Ile Ser Val Val Gly Ser Ser Asp Phe Cys Pro Pro Gly
260 265 270
His Asn Thr Lys Lys Ala Ser Tyr Ser Gly Val Ser Leu Phe Ser Asn
275 280 285
Pro Val Gln Tyr Trp Glu Ile Gln Pro Ser Thr Phe Arg Cys Val Tyr
290 295 300
Val Arg Ser Ala Ile Gln Leu Gly Asn Tyr Lys
305 310 315
<210> 64
<211> 636
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic
<400> 64
Asp Phe Glu Pro Ser Leu Gly Pro Val Cys Pro Phe Arg Cys Gln Cys
1 5 10 15
His Leu Arg Val Val Gln Cys Ser Asp Leu Gly Leu Asp Lys Val Pro
20 25 30
Lys Asp Leu Pro Pro Asp Thr Thr Leu Leu Asp Leu Gln Asn Asn Lys
35 40 45
Ile Thr Glu Ile Lys Asp Gly Asp Phe Lys Asn Leu Lys Asn Leu His
50 55 60
Ala Leu Ile Leu Val Asn Asn Lys Ile Ser Lys Val Ser Pro Gly Ala
65 70 75 80
Phe Thr Pro Leu Val Lys Leu Glu Arg Leu Tyr Leu Ser Lys Asn Gln
85 90 95
Leu Lys Glu Leu Pro Glu Lys Met Pro Lys Thr Leu Gln Glu Leu Arg
100 105 110
Ala His Glu Asn Glu Ile Thr Lys Val Arg Lys Val Thr Phe Asn Gly
115 120 125
Leu Asn Gln Met Ile Val Ile Glu Leu Gly Thr Asn Pro Leu Lys Ser
130 135 140
Ser Gly Ile Glu Asn Gly Ala Phe Gln Gly Met Lys Lys Leu Ser Tyr
145 150 155 160
Ile Arg Ile Ala Asp Thr Asn Ile Thr Ser Ile Pro Gln Gly Leu Pro
165 170 175
Pro Ser Leu Thr Glu Leu His Leu Asp Gly Asn Lys Ile Ser Arg Val
180 185 190
Asp Ala Ala Ser Leu Lys Gly Leu Asn Asn Leu Ala Lys Leu Gly Leu
195 200 205
Ser Phe Asn Ser Ile Ser Ala Val Asp Asn Gly Ser Leu Ala Asn Thr
210 215 220
Pro His Leu Arg Glu Leu His Leu Asp Asn Asn Lys Leu Thr Arg Val
225 230 235 240
Pro Gly Gly Leu Ala Glu His Lys Tyr Ile Gln Val Val Tyr Leu His
245 250 255
Asn Asn Asn Ile Ser Val Val Gly Ser Ser Asp Phe Cys Pro Pro Gly
260 265 270
His Asn Thr Lys Lys Ala Ser Tyr Ser Gly Val Ser Leu Phe Ser Asn
275 280 285
Pro Val Gln Tyr Trp Glu Ile Gln Pro Ser Thr Phe Arg Cys Val Tyr
290 295 300
Val Arg Ser Ala Ile Gln Leu Gly Asn Tyr Lys Ser Gly Gly Gly Gly
305 310 315 320
Ser Asp Phe Glu Pro Ser Leu Gly Pro Val Cys Pro Phe Arg Cys Gln
325 330 335
Cys His Leu Arg Val Val Gln Cys Ser Asp Leu Gly Leu Asp Lys Val
340 345 350
Pro Lys Asp Leu Pro Pro Asp Thr Thr Leu Leu Asp Leu Gln Asn Asn
355 360 365
Lys Ile Thr Glu Ile Lys Asp Gly Asp Phe Lys Asn Leu Lys Asn Leu
370 375 380
His Ala Leu Ile Leu Val Asn Asn Lys Ile Ser Lys Val Ser Pro Gly
385 390 395 400
Ala Phe Thr Pro Leu Val Lys Leu Glu Arg Leu Tyr Leu Ser Lys Asn
405 410 415
Gln Leu Lys Glu Leu Pro Glu Lys Met Pro Lys Thr Leu Gln Glu Leu
420 425 430
Arg Ala His Glu Asn Glu Ile Thr Lys Val Arg Lys Val Thr Phe Asn
435 440 445
Gly Leu Asn Gln Met Ile Val Ile Glu Leu Gly Thr Asn Pro Leu Lys
450 455 460
Ser Ser Gly Ile Glu Asn Gly Ala Phe Gln Gly Met Lys Lys Leu Ser
465 470 475 480
Tyr Ile Arg Ile Ala Asp Thr Asn Ile Thr Ser Ile Pro Gln Gly Leu
485 490 495
Pro Pro Ser Leu Thr Glu Leu His Leu Asp Gly Asn Lys Ile Ser Arg
500 505 510
Val Asp Ala Ala Ser Leu Lys Gly Leu Asn Asn Leu Ala Lys Leu Gly
515 520 525
Leu Ser Phe Asn Ser Ile Ser Ala Val Asp Asn Gly Ser Leu Ala Asn
530 535 540
Thr Pro His Leu Arg Glu Leu His Leu Asp Asn Asn Lys Leu Thr Arg
545 550 555 560
Val Pro Gly Gly Leu Ala Glu His Lys Tyr Ile Gln Val Val Tyr Leu
565 570 575
His Asn Asn Asn Ile Ser Val Val Gly Ser Ser Asp Phe Cys Pro Pro
580 585 590
Gly His Asn Thr Lys Lys Ala Ser Tyr Ser Gly Val Ser Leu Phe Ser
595 600 605
Asn Pro Val Gln Tyr Trp Glu Ile Gln Pro Ser Thr Phe Arg Cys Val
610 615 620
Tyr Val Arg Ser Ala Ile Gln Leu Gly Asn Tyr Lys
625 630 635
<210> 65
<211> 106
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic
<400> 65
Leu Arg Ala His Glu Asn Glu Ile Thr Lys Val Arg Lys Val Thr Phe
1 5 10 15
Asn Gly Leu Asn Gln Met Ile Val Ile Glu Leu Gly Thr Asn Pro Leu
20 25 30
Lys Ser Ser Gly Ile Glu Asn Gly Ala Phe Gln Gly Met Lys Lys Leu
35 40 45
Ser Tyr Ile Arg Ile Ala Asp Thr Asn Ile Thr Ser Ile Pro Gln Gly
50 55 60
Leu Pro Pro Ser Leu Thr Glu Leu His Leu Asp Gly Asn Lys Ile Ser
65 70 75 80
Arg Val Asp Ala Ala Ser Leu Lys Gly Leu Asn Asn Leu Ala Lys Leu
85 90 95
Gly Leu Ser Phe Asn Ser Ile Ser Ala Val
100 105
<210> 66
<211> 218
<212> PRT
<213> Artificial Sequence
<220>
<223> synthetic
<400> 66
Leu Arg Ala His Glu Asn Glu Ile Thr Lys Val Arg Lys Val Thr Phe
1 5 10 15
Asn Gly Leu Asn Gln Met Ile Val Ile Glu Leu Gly Thr Asn Pro Leu
20 25 30
Lys Ser Ser Gly Ile Glu Asn Gly Ala Phe Gln Gly Met Lys Lys Leu
35 40 45
Ser Tyr Ile Arg Ile Ala Asp Thr Asn Ile Thr Ser Ile Pro Gln Gly
50 55 60
Leu Pro Pro Ser Leu Thr Glu Leu His Leu Asp Gly Asn Lys Ile Ser
65 70 75 80
Arg Val Asp Ala Ala Ser Leu Lys Gly Leu Asn Asn Leu Ala Lys Leu
85 90 95
Gly Leu Ser Phe Asn Ser Ile Ser Ala Val Ser Gly Gly Gly Gly Ser
100 105 110
Leu Arg Ala His Glu Asn Glu Ile Thr Lys Val Arg Lys Val Thr Phe
115 120 125
Asn Gly Leu Asn Gln Met Ile Val Ile Glu Leu Gly Thr Asn Pro Leu
130 135 140
Lys Ser Ser Gly Ile Glu Asn Gly Ala Phe Gln Gly Met Lys Lys Leu
145 150 155 160
Ser Tyr Ile Arg Ile Ala Asp Thr Asn Ile Thr Ser Ile Pro Gln Gly
165 170 175
Leu Pro Pro Ser Leu Thr Glu Leu His Leu Asp Gly Asn Lys Ile Ser
180 185 190
Arg Val Asp Ala Ala Ser Leu Lys Gly Leu Asn Asn Leu Ala Lys Leu
195 200 205
Gly Leu Ser Phe Asn Ser Ile Ser Ala Val
210 215
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| БИСПЕЦИФИЧЕСКОЕ АНТИТЕЛО ПРОТИВ ВИРУСА БЕШЕНСТВА И ЕГО ПРИМЕНЕНИЕ | 2019 |
|
RU2764740C1 |
| КОНСТРУКЦИИ СЛИТОГО БЕЛКА ДЛЯ ЗАБОЛЕВАНИЯ, СВЯЗАННОГО С КОМПЛЕМЕНТОМ | 2019 |
|
RU2824402C2 |
| СЛИТЫЙ БЕЛОК, ВКЛЮЧАЮЩИЙ IL-12 И АНТИТЕЛО ПРОТИВ FAP, И ЕГО ПРИМЕНЕНИЕ | 2021 |
|
RU2831612C1 |
| НОВЫЕ СЛИТЫЕ БЕЛКИ, СПЕЦИФИЧЕСКИЕ В ОТНОШЕНИИ CD137 И GPC3 | 2020 |
|
RU2814653C2 |
| АНТИТЕЛО ПРОТИВ ФАКТОРА РОСТА СОЕДИНИТЕЛЬНОЙ ТКАНИ И ЕГО ПРИМЕНЕНИЕ | 2020 |
|
RU2819228C2 |
| НЕВИРУСНЫЙ ИММУНОТАРГЕТИНГ | 2020 |
|
RU2833379C2 |
| ВЫСОКОАФФИННЫЕ АНТИ-PD-1 И АНТИ-LAG-3 АНТИТЕЛА И ПОЛУЧЕННЫЕ ИЗ НИХ БИСПЕЦИФИЧЕСКИЕ СВЯЗЫВАЮЩИЕ БЕЛКИ | 2019 |
|
RU2782381C2 |
| БИСПЕЦИФИЧНЫЙ СЛИТЫЙ БЕЛОК И ЕГО ПРИМЕНЕНИЕ | 2021 |
|
RU2801528C2 |
| Белки, связывающие NKG2D, CD16 и опухолеассоциированный антиген | 2018 |
|
RU2816716C2 |
| АНТИТЕЛА К РЕЦЕПТОРУ IL4 ДЛЯ ПРИМЕНЕНИЯ В ВЕТЕРИНАРИИ | 2019 |
|
RU2837141C2 |
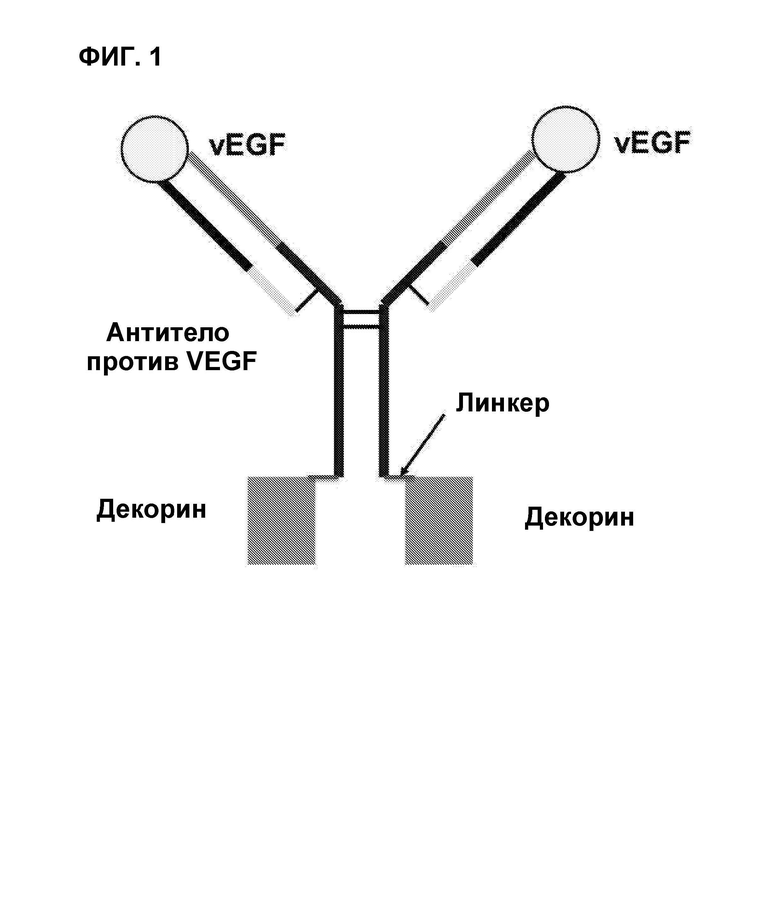
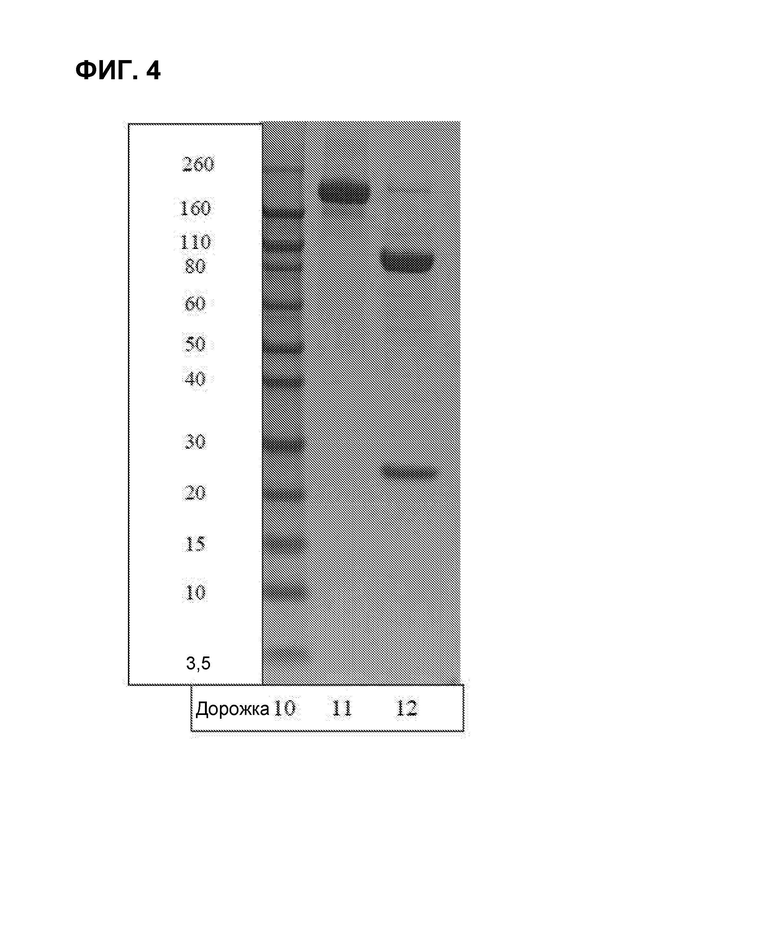

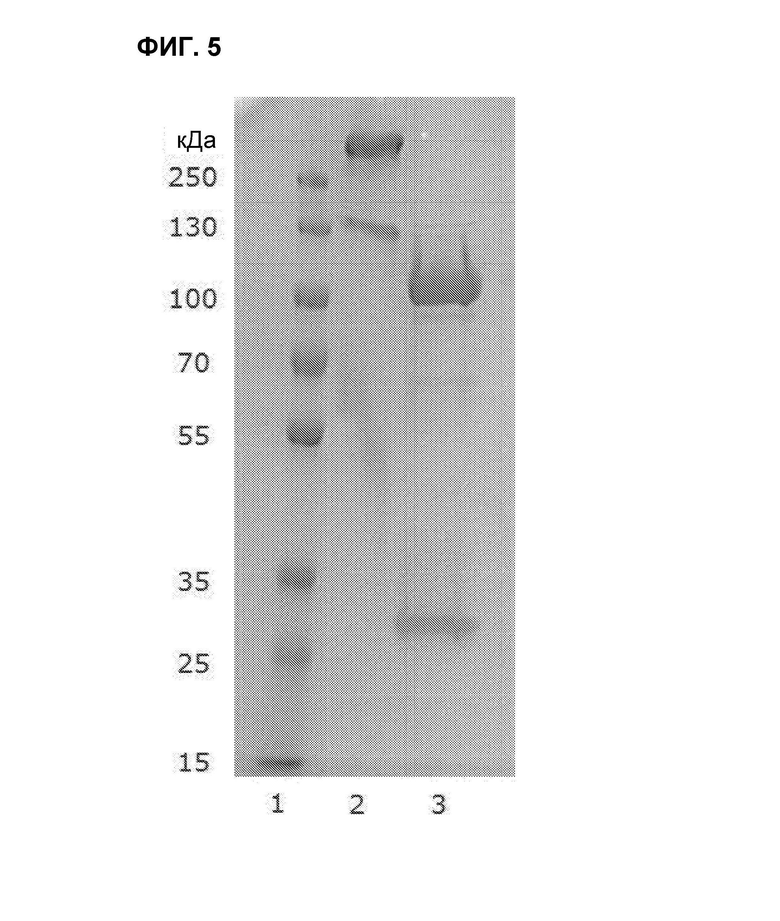
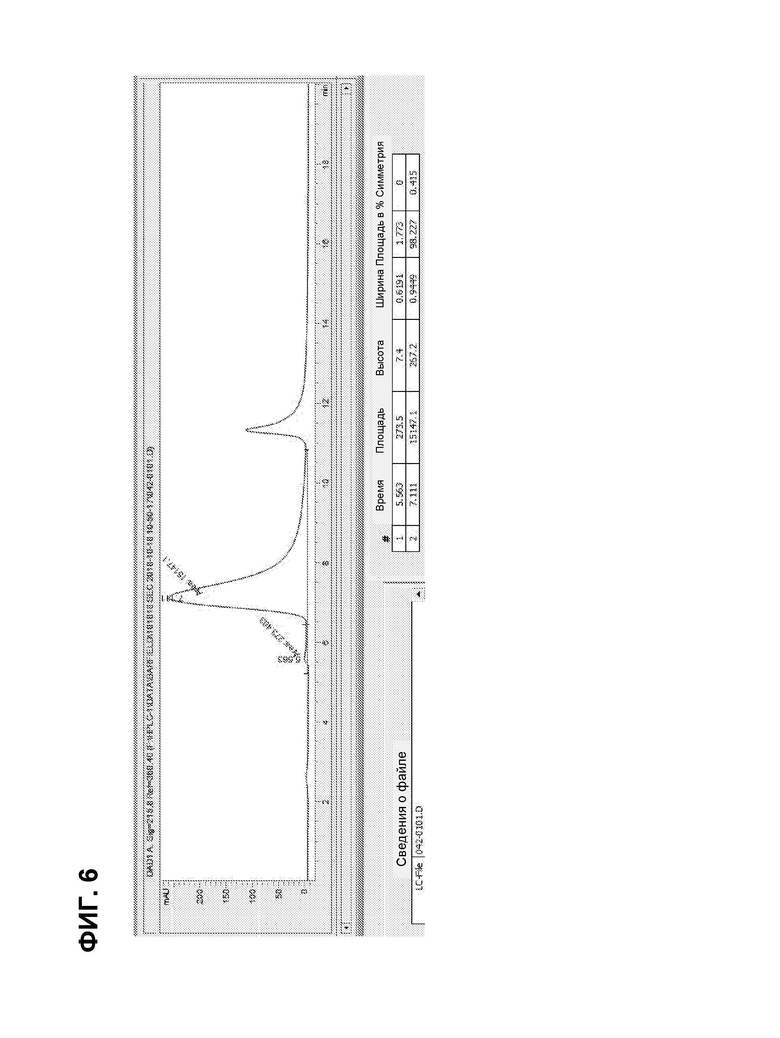
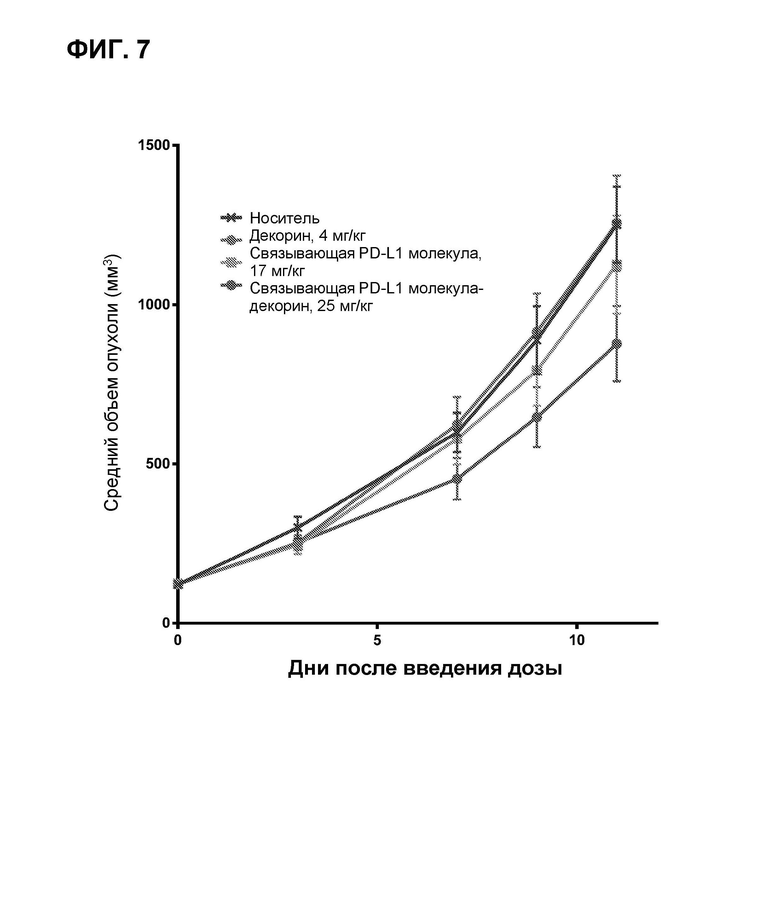
Изобретение относится к биомедицине и биотехнологии, в частности к полифункциональным белковым молекулам, содержащим декорин, и их применениям. В частности, настоящее изобретение относится к полифункциональным белковым молекулам, содержащим декорин и нацеливающий полипептид, такой как антитело, а также способам их получения и их применениям. Изобретение предоставляет новые терапевтические агенты, которые нацелены на VEGF или иммунные контрольные точки. 6 н. и 16 з.п. ф-лы, 7 ил., 4 табл., 3 пр.
1. Белковая молекула для лечения нарушений, характеризующихся ангиогенезом или ростом опухоли, содержащая по меньшей мере одну молекулу декорина, представляющую собой коровый белок декорина, имеющий по меньшей мере 95% идентичность с последовательностью SEQ ID NO: 7, связанный с молекулой антитела, выбранной из группы, состоящей из бевацизумаба, ранибизумаба, ипилимумаба, атезолизумаба, авелумаба, дурвалумаба, ниволумаба и пембролизумаба.
2. Белковая молекула по п. 1, где указанный коровый белок декорина содержит мутацию в положении 4 корового белка зрелого декорина.
3. Белковая молекула по п. 2, где указанная мутация представляет собой мутацию по типу замены серина на аланин, и при этом молекула декорина, содержащая указанную мутацию, представлена в SEQ ID NO: 7.
4. Белковая молекула по любому из пп. 1-3, где указанный белок содержит две или более копий полипептида декорина.
5. Белковая молекула по любому из пп. 1-4, где указанная молекула декорина содержит домен или домены декорина, которые связывают сигнальную молекулу, при этом указанная сигнальная молекула представляет собой трансформирующий фактор роста-β (TGF-β).
6. Белковая молекула по п. 5, где связывающий TGF-β домен содержит последовательность SEQ ID NO: 64 или SEQ ID NO: 66.
7. Белковая молекула по любому из пп. 5 или 6, содержащая две или более копий молекулы декорина.
8. Белковая молекула по любому из пп. 1-7, где указанная молекула декорина функционально связана с тяжелой цепью антитела.
9. Белковая молекула по любому из пп. 1-8, представляющая собой гибридный белок.
10. Белковая молекула по любому из пп. 1-8, где указанная молекула декорина химически связана с антителом.
11. Нуклеиновая кислота, кодирующая белковую молекулу по любому из пп. 1-10.
12. Вектор экспрессии, содержащий нуклеиновую кислоту по п. 11.
13. Клетка-хозяин для экспрессии белковой молекулы по любому из пп. 1-10, содержащая вектор экспрессии по п. 12.
14. Применение белковой молекулы по любому из пп. 1-10, молекулы нуклеиновой кислоты по п. 11 или вектора по п. 12 для ингибирования белка-мишени, выбранного из группы, состоящей из VEGF-A, PD-1, PD-L1, CTLA-4, и сигнальной молекулы в клетке.
15. Применение по п. 14, где указанная клетка представляет собой клетку in vitro или in vivo.
16. Применение по п. 14, где указанная клетка находится в организме субъекта.
17. Применение по любому из пп. 14-16, где указанное приведение в контакт приводит к ингибированию активности, выбранной из группы, состоящей из ангиогенеза, активности PD-1, активности PD-L1, активности CTLA-4, активности PD-L2.
18. Применение по любому из пп. 14-17, где указанные клетки представляют собой раковую клетку, выбранную из группы, состоящей из клеток рака легкого, колоректального рака, рака печени, рака молочной железы, рака почки, рака шейки матки, рака яичника и глиобластомы.
19. Применение по любому из пп. 14-18, где указанный сигнальный белок выбран из группы, состоящей из трансформирующего фактора роста-β (TGF-β), фактора роста соединительной ткани (CTGF), тромбоцитарного фактора роста (PDGF), рецептора 2 фактора роста эндотелия сосудов (VEGFR2), рецептора фактора роста гепатоцитов (HGFR), рецептора инсулиноподобного фактора роста 1 (IGF-1R), различных рецепторов эпидермального фактора роста (EGFR), миостатина и C1q.
20. Применение по любому из пп. 14-19, где указанный сигнальный белок представляет собой трансформирующий фактор роста-β (TGF-β).
21. Применение белковой молекулы по любому из пп. 1-10, молекулы нуклеиновой кислоты по п. 11 или вектора по п. 12 при лечении нарушений, характеризующихся ангиогенезом или ростом опухоли.
22. Применение по п. 21, где указанная опухоль выбрана из группы, состоящей из рака легкого, колоректального рака, рака печени, рака молочной железы, рака почки, рака шейки матки, рака яичника и глиобластомы.
| СПОСОБ ПРОИЗВОДСТВА КОНСЕРВОВ "ТРЕСКА ТУШЕНАЯ С КАПУСТОЙ" | 2013 |
|
RU2506862C1 |
| WO 2017214706 A1, 21.12.2017 | |||
| Способ создания силы и устройство для его осуществления | 1989 |
|
SU1789424A1 |
| JARVINEN T.A., RUOSLAHTI E | |||
| Target-seeking antifibrotic compound enhances wound healing and suppresses scar formation in mice, Proc Natl Acad Sci USA, 2010, vol | |||
| Счетный сектор | 1919 |
|
SU107A1 |
| Устройство для выпрямления многофазного тока | 1923 |
|
SU50A1 |
| Газовый огнетушитель для кинопроектора | 1930 |
|
SU21671A1 |
| KHAN G.A | |||
| et al | |||
| Аппарат для очищения воды при помощи химических реактивов | 1917 |
|
SU2A1 |
Авторы
Даты
2024-07-22—Публикация
2019-06-28—Подача