Перекрестная ссылка на родственную заявку
Настоящая заявка претендует на приоритет по предварительной заявке на патент США No. 62/381,441, поданной 30 августа 2016 г., которая включена сюда путем ссылки во всей полноте.
Область техники, к которой относится изобретение
Настоящее изобретение касается микробиологических композиций и способов их применения, в особенности для сельскохозяйственных процессов и применений.
Уровень техники
Вследствие роста населения потребление продуктов питания также возрастает. С другой стороны, значительно уменьшаются возделываемые сельскохозяйственные угодья и продуктивность из-за глобальной индустриализации, засухи, засоления и глобального потепления (Galamero et al., In: Microbial Strategies for Crop Improvement, Springer Berlin, pp. 1-22, 2009). Эта проблема может быть решена путем ведения устойчивого сельского хозяйства, основным принципом которого является значительное снижение химических веществ типа удобрений, инсектицидов и гербицидов при одновременном уменьшении выброса парниковых газов.
Чрезмерное применение химических удобрений в сельском хозяйстве приводит к большому числу экологических проблем, так как некоторые удобрения содержат тяжелые металлы (например, кадмий и хром) и высокие концентрации радионуклидов. Эти удобрения в агроэкосистеме являются основным источником тяжелых металлов и радионуклидов у растений, а некоторые ведут к накоплению неорганических загрязнений (Savci, Int. J. Env. Sci. Dev. 3: 77-80, 2012; Malakoff, Science 281: 190-192, 1998). В теплицах и аквакультурах используется особенно большое количество химических удобрений на пике сезона, что ведет к загрязнению водных ресурсов, а урожайность и качество продукции ухудшается. В свете этих недостатков химических удобрений представляются перспективными полезные для растений микробиологические инокулянты в качестве компонентов комплексной стратегии управления питательными веществами.
Сущность изобретения
Здесь раскрыты композиции, включающие клетки из определенной группы видов микроорганизмов (к примеру, 3 видов, 16 видов, 17 видов, 19 видов, 20 видов, 21 вида или 22 видов микроорганизмов). В некоторых воплощениях композиции включают клетки из одного или нескольких видов микроорганизмов, обладающие такими функциональными характеристиками или активностями (типа метаболической активности), без ограничения, как азотный метаболизм, солеустойчивость, активность солюбилизации солей фосфата и/или кальция и/или цинка, целлюлолитическая активность, хитинолитическая активность, продукция фитогормонов, активность метаболизма железа и/или дефосфорилирования органических веществ. В некоторых воплощениях композиции включают клетки из одного или нескольких (как-то 2 или больше, 3 или больше, 4 или больше, 5 или больше, 6 или больше, 7 или больше, 8 или больше, 9 или больше, 10 или больше, 11 или больше, 12 или больше, 13 или больше, 14 или больше, 15 или больше) видов микроорганизмов, которые растут в аэробных условиях. В других воплощениях композиции включают клетки из одного или нескольких (как-то 2 или больше, 3 или больше, 4 или больше, 5 или больше) видов микроорганизмов, которые растут в анаэробных условиях. В одном неограничительном примере композиция включает клетки из 16 видов микроорганизмов, растущих в аэробных условиях, и клетки 6 видов микроорганизмов, растущих в анаэробных условиях. Аэробные и анаэробные условия роста включают, без ограничения, условия, описанные здесь в Примере 1.
В одном воплощении композиция включает клетки таких видов микроорганизмов, как Lactobacillus delbrueckii, Virgibacillus halophilus, Azotobacter vinelandii, Clostridium pasteurianum, Paenibacillus chibensis, Streptomyces griseus, Pseudomonas sp. (близкородственных P.entomophila, P. fluorescens и P. putida, к примеру, таких видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 17), Pseudomonas putida, Bacillus sp. (близкородственных B. kochii, B. pocheonensis и Bacillus sp. штамма R-27341, к примеру, таких видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 15), Bacillus amyloliquefaciens, Oceanobacillus oncorhynchi, Paenibacillus lautus (например, близкородственных Paenibacillus lautus и Paenibacillus sp. штамма Y412MC10, к примеру, таких видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 12), Bacillus licheniformis, Lactobacillus vini, Paenibacillus cookii, Bacillus subtilis, Lactobacillus buchneri, Bacillus megaterium, Acetobacter pasteurianus, Clostridium beijerinckii, Lactobacillus casei/paracasei и Bacillus flexus. В другом воплощении композиция включает клетки таких видов микроорганизмов, как Lactobacillus delbrueckii, Virgibacillus halophilus, Azotobacter vinelandii, Clostridium pasteurianum, Streptomyces griseus, Pseudomonas sp. (близкородственных P. entomophila, P. fluorescens и P. putida, к примеру, таких видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 17), Pseudomonas putida, Bacillus sp. (близкородственных B. kochii, B. pocheonensis и Bacillus sp. штамма R-27341, к примеру, таких видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 15), Bacillus amyloliquefaciens, Oceanobacillus oncorhynchi, Bacillus licheniformis, Lactobacillus vini, Bacillus subtilis, Lactobacillus buchneri, Bacillus megaterium, Acetobacter pasteurianus, Clostridium beijerinckii, Lactobacillus casei/paracasei и Bacillus flexus. В других воплощениях композиции включают клетки таких видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности каждой из SEQ ID NO: 3-24; каждой из SEQ ID NO: 3-8, 10, 11, 13-18 и 20-24; каждой из SEQ ID NO: 3-7 и 9-24; каждой из SEQ ID NO: 3-7, 10, 11, 13-18 и 20-24; каждой из SEQ ID NO: 3-8, 10, 11, 14, 16-18, 20-22 и 24; или каждой из SEQ ID NO: 3-14, 16-22 и 24. В другом воплощении композиция включает коллекцию микроорганизмов, содержащихся в Американской коллекции типовых культур под депозитным номером PTA-123288, PTA-123298 и/или PTA-123289.
В других воплощениях композиция включает клетки из по меньшей мере одного вида микроорганизмов с активностью азотного метаболизма, по меньшей мере одного вида микроорганизмов с устойчивостью к 5% NaCl, по меньшей мере одного вида микроорганизмов с активностью солюбилизации солей фосфата и/или кальция и/или цинка, по меньшей мере одного вида микроорганизмов с целлюлолитической и/или хитинолитической активностью, по меньшей мере одного вида микроорганизмов с активностью метаболизма яблочной кислоты, по меньшей мере одного вида микроорганизмов с активностью продукции фитогормонов (типа индола (ауксина)), по меньшей мере одного вида микроорганизмов с активностью метаболизма железа, по меньшей мере одного вида микроорганизмов с активностью дефосфорилирования органических веществ либо комбинации каких-либо двух или нескольких из них.
Также здесь раскрыты способы применения представленных композиций, которые включают обработку почвы, растений, частей растений или семян данной композицией. Микробиологические композиции можно наносить на почву, растения, части растений и/или семена по отдельности или в комбинации с другими компонентами (типа хитина, хитозана, глюкозамина, аминокислот и/или жидких удобрений).
В других воплощениях представленные микробиологические композиции применяются в способах разложения биологических материалов типа содержащих хитин биологических материалов. В некоторых примерах содержащие хитин материалы смешивают с приведенной микробиологической композицией и подвергают ферментации с получением ферментированной смеси. Ферментированная смесь необязательно может быть разделена на твердую и жидкую фракции. Эти фракции впоследствии могут применяться в сельском хозяйстве в сочетании с представленными микробиологическими композициями или же в дальнейших процессах разложения.
Вышеизложенные и другие особенности изобретения станут более понятными из следующего подробного описания, которое приводится с привлечением сопровождающих фигур.
Краткое описание фигур
На фиг. 1 представлена схема типичного процесса биологического разложения содержащего хитин биологического материала (в качестве примера – отходов от креветок) с помощью микробиологической композиции.
На фиг. 2 представлена диаграмма индекса листовой поверхности (LAI) на 33-й день у концов настоящих листьев растений огурцов при указанной обработке.
На фиг. 3 представлена диаграмма сухого веса у 17-дневных ростков кукурузы при указанной обработке.
На фиг. 4 представлена диаграмма урожайности томатов (зеленые и красные помидоры) при указанной обработке.
На фиг. 5 представлена диаграмма урожайности кукурузы при указанной обработке.
На фиг. 6 представлена диаграмма общего урожая капусты при указанной обработке.
Перечень последовательностей
Нуклеотидные и аминокислотные последовательности, перечисленные здесь или в прилагаемом списке последовательностей, приводятся с использованием стандартных буквенных сокращений для оснований нуклеотидов и аминокислот, как определено в 37 C.F.R. § 1.822. По крайней мере в некоторых случаях приводится только одна нить каждой последовательности нуклеиновой кислоты, но при любой ссылке на приведенную нить считается включенной и комплементарная нить.
SEQ ID NO: 1 и 2 – нуклеотидные последовательности прямого и обратного праймеров для 16S-рДНК, соответственно.
SEQ ID NO: 3 – нуклеотидная последовательность 16S-рДНК из микроорганизма Bacillus megaterium.
SEQ ID NO: 4 – нуклеотидная последовательность 16S-рДНК из микроорганизма Lactobacillus casei/paracasei.
SEQ ID NO: 5 – нуклеотидная последовательность 16S-рДНК из микроорганизма Clostridium beijerinckii.
SEQ ID NO: 6 – нуклеотидная последовательность 16S-рДНК из микроорганизма Acetobacter pasteurianus.
SEQ ID NO: 7 – нуклеотидная последовательность 16S-рДНК из микроорганизма Lactobacillus buchneri.
SEQ ID NO: 8 – нуклеотидная последовательность 16S-рДНК из микроорганизма Bacillus subtilis.
SEQ ID NO: 9 – нуклеотидная последовательность 16S-рДНК из микроорганизма Paenibacillus cookii.
SEQ ID NO: 10 – нуклеотидная последовательность 16S-рДНК из микроорганизма Lactobacillus vini.
SEQ ID NO: 11 – нуклеотидная последовательность 16S-рДНК из микроорганизма Bacillus licheniformis.
SEQ ID NO: 12 – нуклеотидная последовательность 16S-рДНК из микроорганизма Paenibacillus lautus.
SEQ ID NO: 13 – нуклеотидная последовательность 16S-рДНК из микроорганизма Oceanobacillus oncorhynchi.
SEQ ID NO: 14 – нуклеотидная последовательность 16S-рДНК из микроорганизма Bacillus amyloliquefaciens.
SEQ ID NO: 15 – нуклеотидная последовательность 16S-рДНК из микроорганизма Bacillus sp.
SEQ ID NO: 16 – нуклеотидная последовательность 16S-рДНК из микроорганизма Pseudomonas putida.
SEQ ID NO: 17 – нуклеотидная последовательность 16S-рДНК из микроорганизма Pseudomonas sp.
SEQ ID NO: 18 – нуклеотидная последовательность 16S-рДНК из микроорганизма Streptomyces griseus.
SEQ ID NO: 19 – нуклеотидная последовательность 16S-рДНК из микроорганизма Paenibacillus chibensis.
SEQ ID NO: 20 – нуклеотидная последовательность 16S-рДНК из микроорганизма Bacillus flexus.
SEQ ID NO: 21 – нуклеотидная последовательность 16S-рДНК из микроорганизма Clostridium pasteurianum.
SEQ ID NO: 22 – нуклеотидная последовательность 16S-рДНК из микроорганизма Azotobacter vinelandii.
SEQ ID NO: 23 – нуклеотидная последовательность 16S-рДНК из микроорганизма Virgibacillus halophilus.
SEQ ID NO: 24 – нуклеотидная последовательность 16S-рДНК из микроорганизма Lactobacillus delbrueckii.
SEQ ID NO: 25-66 и 69-136 – нуклеотидные последовательности видоспецифичных олигонуклеотидных праймеров и зондов.
SEQ ID NO: 67-68 – нуклеотидные последовательности универсальных праймеров для прокариотической 16S-рРНК.
Раскрытие сущности изобретения
I Термины
Если не указано иначе, технические термины применяются в соответствии со стандартным употреблением. Определения распространенных терминов в молекулярной биологии приведены в Krebs et al., Lewin’s Genes XI, Jones and Bartlett Learning, 2012 (ISBN 1449659853); Kendrew et al. (eds.), The Encyclopedia of Molecular Biology, Blackwell Publishers, 1994 (ISBN 0632021829); Robert A. Meyers (ed.), Molecular Biology and Biotechnology: a Comprehensive Desk Reference, John Wiley & Sons, Inc., 2011 (ISBN 8126531789); и George P. Rédei, Encyclopedic Dictionary of Genetics, Genomics, and Proteomics, 2nd Edition, 2003 (ISBN: 0-471-26821-6).
Следующие объяснения терминов и способов приводятся для лучшего описания настоящего изобретения и для руководства рядовых специалистов в данной области при практическом применения настоящего изобретения. Формы единственного числа означают одно или несколько, если контекстом явно не предписано иное. К примеру, термин “содержащий клетку” включает в себя одну или несколько клеток и считается эквивалентным фразе “содержащий по меньшей мере одну клетку”. В настоящем изобретении термин “содержит” означает “включает”. Таким образом, “содержащий A или B” означает “включающий A, B либо A и B”, не исключая дополнительные элементы. Все публикации, патентные заявки, патенты и другие приведенные здесь ссылки включены сюда путем ссылки во всей полноте для всех целей. В случае противоречия следует руководствоваться настоящим описанием, включая пояснения терминов.
Подходящие для практического применения или тестирования изложенной технологии методы и материалы описаны ниже, хотя в этих же целях можно использовать методы и материалы, подобные или эквивалентные тем, что описаны здесь. Материалы, методы и примеры приводятся только для иллюстрации и не предназначены для ограничения.
Для облегчения изложения различных воплощений настоящего изобретения приводятся следующие пояснения определенных терминов.
Водное животное – животное, которое живет в соленой или пресной воде. В определенных воплощениях, приведенных здесь, водные животные включают водных членистоногих, как-то креветки, криль, веслоногие, ракушки, крабы, омары и раки. В других воплощениях водные животные включает рыб. Побочные продукты водных животных включают любые части водных животных, в особенности части, получаемые при промышленной переработке водных животных. Так, в некоторых примерах побочные продукты водных животных включают одно или несколько из цефалоторакса или экзоскелета креветок, экзоскелета крабов или омаров либо кожи или чешуи рыб.
Контактирование – приведение в непосредственное физическое соприкосновение, в том числе в твердой и жидкой форме. Например, контактирование может происходить между одним или несколькими микроорганизмами (типа микроорганизмов в микробной консорции) и биологическим образцом в растворе. Контактирование также может происходить между одним или несколькими микроорганизмами (типа микроорганизмов в микробной консорции) и почвой, растениями и/или частями растений (типа листвы, стеблей, побегов, корней и/или семян).
Культуральная среда – набор условий культивирования (который в некоторых примерах является синтетическим или не встречающимся в природе), включающий питательные вещества для поддержания жизнеспособности, функционирования и/или роста определенной популяции клеток типа одного или нескольких видов микроорганизмов. Обычно культуральные среды включают такие компоненты, как источник углерода, источник азота и буфер для поддержания рН. Дополнительные компоненты в культуральных средах также могут включать один или несколько гормонов, факторов роста, ингибиторов протеаз, гидролизатов белка, протекторов от разрыва, белков, витаминов, микроэлементов, неорганических солей, минералов и/или липидов.
Культивирование – целенаправленное выращивание одного или нескольких организмов или клеток в присутствии усваиваемых источников углерода, азота и минеральных солей. В одном примере такое выращивание может происходить в твердой или полутвердой питательной среде или в жидкой среде, в которой растворены или суспендированы питательные вещества. В другом примере культивирование может протекать на поверхности или в погруженной культуре. Питательная среда может состоять из сложных питательных веществ или может быть химически определенной.
Ферментация – процесс, который ведет к расщеплению сложных органических соединений на более простые соединения, к примеру, клетками микроорганизмов (типа бактерий и/или грибков). Процесс ферментации может протекать в аэробных условиях, анаэробных условиях или в тех и других (к примеру, в большом объеме, где некоторые порции являются аэробными, а другие – анаэробными). В некоторых неограничительных воплощениях ферментация включает ферментативное и/или неферментативное расщепление соединений, присутствующих у водных животных или в побочных продуктах от животных типа хитина.
Выделенный – “выделенный” биологический компонент (типа нуклеиновой кислоты, белка или организма) существенно отделен или очищен от других биологических компонентов (типа других клеток, остатков клеток или других белков или нуклеиновых кислот). Биологические компоненты, которые были “выделены”, включают компоненты, очищенные стандартными методами очистки. Термин также охватывает рекомбинантные нуклеиновые кислоты, белки или микроорганизмы, а также химически синтезированные нуклеиновые кислоты или пептиды. Термин “выделенный” (либо “обогащенный” или “очищенный”) не требует абсолютной чистоты и может включать микроорганизмы или молекулы, которые выделены по меньшей мере на 50%, как-то по меньшей мере на 75%, 80%, 90%, 95% 98%, 99% или даже на 100%.
Жидкое удобрение – водный раствор или суспензия, содержащая растворимый азот. В некоторых примерах растворимый азот в жидком удобрении включает органический источник азота типа мочевины или мочевины, полученной из безводного аммиака (как-то раствор мочевины и нитрата аммония (UAN)). Также можно использовать водный аммиак (20-32% безводного аммиака). В других примерах растворимый азот в жидком удобрении включает азотсодержащие неорганические соли типа гидроксида аммония, нитрата аммония, сульфата аммония, пирофосфата аммония, тиосульфата аммония или комбинации из двух или нескольких из них. В некоторых воплощениях жидкое удобрение включает не встречающийся в природе источник азота (типа пирофосфата аммония или тиосульфата аммония) и/или другие не встречающиеся в природе компоненты.
Обычные жидкие неприродные смеси удобрений определяются по содержанию в них азота-фосфата-калия (содержанию N-P-K) и включают добавление и других компонентов типа серы или цинка. Примеры искусственных смесей включают 10-34-0, 10-30-0 с 2% серы и 0,25% цинка (хелатного), 11-37-0, 12-30-0 с 3% серы, 2-4-12, 2-6-12, 4-10-10, 3-18-6, 7-22-5, 8-25-3, 15-15-3, 17-17-0 с 2% серы, 18-18-0, 18-18-0 с 2% серы, 28-0-0 UAN, 9-27-0 с 2% серы и тиосульфатом калия.
Микробы – микроорганизмы, включая, без ограничения, бактерии, архебактерии, грибки и водоросли (как-то микроводоросли). В некоторых примерах микробы представляют собой одноклеточные организмы (к примеру, бактерии, цианобактерии, некоторые грибы или некоторые водоросли). В других примерах термин микробы включает многоклеточные организмы типа определенных грибков или водорослей (к примеру, многоклеточные мицелиальные грибы или многоклеточные водоросли).
Микробиологическая (микробная) композиция – композиция (которая может быть твердой, жидкой или по крайней мере частично той и другой), которая включает клетки по меньшей мере одного типа (или вида) микроорганизмов (или популяцию клеток по меньшей мере одного типа микроорганизмов). В некоторых примерах микробиологическая композиция включает клетки одного или нескольких типов (видов) микроорганизмов (либо одной или нескольких популяций микроорганизмов) в жидкой среде (типа среды для хранения, культивирования или ферментации), к примеру, в виде суспензии в жидкой среде. В других примерах микробиологическая композиция включает клетки одного или нескольких типов (видов) микроорганизмов (либо одной или нескольких популяций микроорганизмов) на поверхности или внутри твердой или студенистой среды (включая, без ограничения, культуральные чашки) либо кашицы или пасты.
Микробный консорциум – смесь, ассоциация или совокупность клеток двух или нескольких видов микроорганизмов, которые в некоторых случаях находятся в физическом контакте друг с другом. Микроорганизмы в консорциуме могут воздействовать друг на друга при непосредственном физическом контакте, посредством биохимических взаимодействий или тем и другим. Например, микроорганизмы в консорциуме могут обмениваться питательными веществами, метаболитами или газами друг с другом. Так, в некоторых примерах по меньшей мере некоторые из микроорганизмов в консорциуме метаболически взаимосвязаны. Такие взаимосвязи могут меняться по своему характеру и степени во времени и при изменении условий культивирования.
II Микробиологические композиции
Предусмотрены микробиологические композиции, включающие клетки из определенной группы микроорганизмов или видов микроорганизмов. В некоторых воплощениях представленные композиции включают клетки из определенной группы микроорганизмов (к примеру, 3 видов, 16 видов, 19 видов, 20 видов, 21 вида или 22 видов микроорганизмов), как указано ниже. Представленные композиции также могут включать в себя один или несколько немикробных компонентов, включая, без ограничения, один или несколько источников углерода, источников азота, буферов, гормонов, факторов роста, ингибиторов протеаз, гидролизатов белков, протекторов от разрыва, белков, аминокислот, витаминов, микроэлементов, неорганических солей, минералов и/или липидов. В некоторых примерах в композиции также могут быть добавлены один или несколько дополнительных видов микроорганизмов, к примеру, для обеспечения или восполнения требуемой активности в композиции.
A. Определенные микробиологические композиции
Предусмотрены композиции, включающие клетки из определенной группы видов микроорганизмов. Например, в некоторых воплощениях композиции включают изоляты микроорганизмов, которые объединены в одну композицию, а в некоторых примерах они совместно культивируются или ферментируются.
В одном примере композиция включает клетки таких видов микроорганизмов, как Lactobacillus delbrueckii, Virgibacillus halophilus, Azotobacter vinelandii, Clostridium pasteurianum, Paenibacillus chibensis, Streptomyces griseus, Pseudomonas sp. (близкородственных P.entomophila, P. fluorescens и P. putida, к примеру, таких видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 17), Pseudomonas putida, Bacillus sp. (близкородственных B. kochii, B. pocheonensis и Bacillus sp. штамма R-27341, к примеру, таких видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 15), Bacillus amyloliquefaciens, Oceanobacillus oncorhynchi, Paenibacillus lautus (например, близкородственных Paenibacillus lautus и Paenibacillus sp. штамма Y412MC10, к примеру, таких видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 12), Bacillus licheniformis, Lactobacillus vini, Paenibacillus cookii, Bacillus subtilis, Lactobacillus buchneri, Bacillus megaterium, Acetobacter pasteurianus, Clostridium beijerinckii, Lactobacillus casei/paracasei и Bacillus flexus. В другом примере композиция включает клетки таких видов микроорганизмов, как Lactobacillus delbrueckii, Virgibacillus halophilus, Azotobacter vinelandii, Clostridium pasteurianum, Streptomyces griseus, Pseudomonas sp. (близкородственных P. entomophila, P. fluorescens и P. putida, к примеру, таких видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 17), Pseudomonas putida, Bacillus sp. (близкородственных B. kochii, B. pocheonensis и Bacillus sp. штамма R-27341, к примеру, таких видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 15), Bacillus amyloliquefaciens, Oceanobacillus oncorhynchi, Bacillus licheniformis, Lactobacillus vini, Bacillus subtilis, Lactobacillus buchneri, Bacillus megaterium, Acetobacter pasteurianus, Clostridium beijerinckii, Lactobacillus casei/paracasei и Bacillus flexus. А еще в одном примере композиция включает клетки таких видов микроорганизмов, как Lactobacillus delbrueckii, Virgibacillus halophilus, Azotobacter vinelandii, Clostridium pasteurianum, Paenibacillus chibensis, Streptomyces griseus, Pseudomonas sp. (близкородственных P. entomophila, P. fluorescens и P. putida, к примеру, таких видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 17), Pseudomonas putida, Bacillus sp. (близкородственных B. kochii, B. pocheonensis и Bacillus sp. штамма R-27341, к примеру, таких видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 15), Bacillus amyloliquefaciens, Oceanobacillus oncorhynchi, Paenibacillus lautus (например, близкородственных Paenibacillus lautus и Paenibacillus sp. штамма Y412MC10, к примеру, таких видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 12), Bacillus licheniformis, Lactobacillus vini, Paenibacillus cookii, Lactobacillus buchneri, Bacillus megaterium, Acetobacter pasteurianus, Clostridium beijerinckii, Lactobacillus casei/paracasei и Bacillus flexus. В следующем примере композиция включает клетки таких видов микроорганизмов, как Lactobacillus delbrueckii, Virgibacillus halophilus, Azotobacter vinelandii, Clostridium pasteurianum, Streptomyces griseus, Pseudomonas sp. (близкородственных P. entomophila, P. fluorescens и P. putida, к примеру, таких видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 17), Pseudomonas putida, Bacillus sp. (близкородственных B. kochii, B. pocheonensis и Bacillus sp. штамма R-27341, к примеру, таких видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 15), Bacillus amyloliquefaciens, Oceanobacillus oncorhynchi, Bacillus licheniformis, Lactobacillus vini, Bacillus subtilis, Lactobacillus buchneri, Bacillus megaterium, Acetobacter pasteurianus, Clostridium beijerinckii, Lactobacillus casei/paracasei и Bacillus flexus.
В других примерах композиция включает клетки таких видов микроорганизмов, как Lactobacillus delbrueckii, Virgibacillus halophilus, Azotobacter vinelandii, Clostridium pasteurianum, Paenibacillus chibensis, Pseudomonas sp. (близкородственных P. entomophila, P. fluorescens и P. putida, к примеру, таких видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 17), Pseudomonas putida, Bacillus sp. (близкородственных B. kochii, B. pocheonensis и Bacillus sp. штамма R-27341, к примеру, таких видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 15), Bacillus amyloliquefaciens, Oceanobacillus oncorhynchi, Paenibacillus lautus (например, близкородственных Paenibacillus lautus и Paenibacillus sp. штамма Y412MC10, к примеру, таких видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 12), Bacillus licheniformis, Lactobacillus vini, Paenibacillus cookii, Bacillus subtilis, Lactobacillus buchneri, Bacillus megaterium, Acetobacter pasteurianus и Lactobacillus casei/paracasei. Еще в одних примерах композиция включает клетки таких видов микроорганизмов, как Lactobacillus delbrueckii, Virgibacillus halophilus, Azotobacter vinelandii, Clostridium pasteurianum, Pseudomonas sp. (близкородственных P. entomophila, P. fluorescens и P. putida, к примеру, таких видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 17), Pseudomonas putida, Bacillus sp. (близкородственных B. kochii, B. pocheonensis и Bacillus sp. штамма R-27341, к примеру, таких видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 15), Bacillus amyloliquefaciens, Oceanobacillus oncorhynchi, Bacillus licheniformis, Lactobacillus vini, Bacillus subtilis, Lactobacillus buchneri, Bacillus megaterium, Acetobacter pasteurianu и Lactobacillus casei/paracasei.
В следующих примерах композиция включает клетки таких видов микроорганизмов, как Lactobacillus delbrueckii, Azotobacter vinelandii, Clostridium pasteurianum, Streptomyces griseus, Pseudomonas sp. (близкородственных P. entomophila, P. fluorescens и P. putida, к примеру, таких видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 17), Pseudomonas putida, Bacillus amyloliquefaciens, Bacillus licheniformis, Lactobacillus vini, Bacillus subtilis, Lactobacillus buchneri, Bacillus megaterium, Acetobacter pasteurianus, Clostridium beijerinckii, Lactobacillus casei/paracasei и Bacillus flexus.
В другом примере композиция включает клетки таких видов микроорганизмов, как Lactobacillus delbrueckii, Azotobacter vinelandii, Clostridium pasteurianum, Paenibacillus chibensis, Streptomyces griseus, Pseudomonas sp. (близкородственных P. entomophila, P. fluorescens и P. putida, к примеру, таких видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 17), Pseudomonas putida, Bacillus amyloliquefaciens, Oceanobacillus oncorhynchi, Paenibacillus lautus (например, близкородственных Paenibacillus lautus и Paenibacillus sp. штамма Y412MC10, к примеру, таких видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 12), Bacillus licheniformis, Lactobacillus vini, Paenibacillus cookii, Bacillus subtilis, Lactobacillus buchneri, Bacillus megaterium, Acetobacter pasteurianus, Clostridium beijerinckii, Lactobacillus casei/paracasei и Bacillus flexus.
В следующем примере композиция включает клетки таких видов микроорганизмов, как Clostridium beijerinckii, Streptomyces griseus и Bacillus flexus.
В определенных примерах используются комбинации этих композиций для получения представленных композиций, к примеру, путем смешивания и совместной ферментации двух композиций.
Рядовым специалистам должно быть известно, что идентификация микроорганизмов, особенно на уровне вида или штамма, не всегда возможна. Как описано в примере 1, микроорганизмы в описанных здесь композициях анализировали путем секвенирования 16S-рДНК и полногеномного секвенирования с последующим сравнением с последовательностями в общедоступных базах данных. Однако из-за ограниченности информации в базах данных по последовательностям (включая мало или никакой информации по некоторым видам или штаммам и/или изменения в номенклатуре с течением времени) может оказаться затруднительным получение идентификации определенных видов или штаммов. Так, в некоторых воплощениях виды микроорганизмов, включенные в представленные композиции, идентифицируются по идентичности их последовательностей с приведенными здесь последовательностями 16S-рДНК (SEQ ID NOs: 3-24).
В некоторых примерах композиция включает клетки таких видов микроорганизмов, у которых последовательности 16S-рДНК по меньшей мере на 95% (как-то по меньшей мере на 96%, на 97%, на 98%, на 99% или даже на 100%) идентичны последовательности 16S-рДНК каждой из SEQ ID NOs: 3-24. В других примерах композиция включает клетки таких видов микроорганизмов, у которых последовательности 16S-рДНК по меньшей мере на 95% (как-то по меньшей мере на 96%, на 97%, на 98%, на 99% или даже на 100%) идентичны последовательностям 16S-рДНК каждой из SEQ ID NOs: 3-8, 10, 11, 13-18 и 20-24. А еще в одних примерах композиция включает клетки таких видов микроорганизмов, у которых последовательности 16S-рДНК по меньшей мере на 95% (как-то по меньшей мере на 96%, на 97%, на 98%, на 99% или даже на 100%) идентичны последовательностям 16S-рДНК каждой из SEQ ID NOs: 3-7 и 9-24, или же клетки таких видов микроорганизмов, у которых последовательности 16S-рДНК по меньшей мере на 95% (как-то по меньшей мере на 96%, на 97%, на 98%, на 99% или даже на 100%) идентичны последовательностям 16S-рДНК каждой из SEQ ID NOs: 3-7, 10, 11, 13-18 и 20-24. В других примерах композиция включает клетки таких видов микроорганизмов, у которых последовательности 16S-рДНК по меньшей мере на 95% (как-то по меньшей мере на 96%, на 97%, на 98%, на 99% или даже на 100%) идентичны последовательностям 16S-рДНК каждой из SEQ ID NOs: 3-8, 10, 11, 14, 16-18, 20-22 и 24, или же клетки таких видов микроорганизмов, у которых последовательности 16S-рДНК по меньшей мере на 95% (как-то по меньшей мере на 96%, на 97%, на 98%, на 99% или даже на 100%) идентичны последовательностям 16S-рДНК каждой из SEQ ID NOs: 3-14, 16-22 и 24.
В некоторых воплощениях композиция включает клетки таких видов микроорганизмов, которые входят в коллекцию микроорганизмов, депонированную в Американской коллекции типовых культур (ATCC, Manassas, VA) от 1 июля 2016 г. под депозитным номером PTA-123288, PTA-123298 или PTA-123289. В некоторых примерах композиция включает клетки таких видов микроорганизмов, которые входят в два или несколько из приведенных депозитов ATCC, к примеру, микроорганизмов под депозитными номерами PTA-123288 и PTA-123289 в ATCC или микроорганизмов под депозитными номерами PTA-123298 и PTA-123289 в ATCC.
В дополнительных воплощениях композиция включает клетки из комбинации видов микроорганизмов, обеспечивающих желательные метаболические характеристики или активности (к примеру, одну или несколько активностей, способствующих росту растений). Так, в некоторых примерах композиция включает клетки по меньшей мере одного вида микроорганизмов с активностью азотного метаболизма (типа денитрификации, фиксации азота и/или продукции уреазы), клетки по меньшей мере одного вида микроорганизмов с устойчивостью к соли (к примеру, рост через 72 часа в среде, содержащей 1%, 2,5%, 5%, 7,5% или 10% соли), клетки по меньшей мере одного вида микроорганизмов с активностью солюбилизации солей фосфата и/или кальция и/или цинка, клетки по меньшей мере одного вида микроорганизмов с целлюлолитической и/или хитинолитической активностью (типа деградации GlcNAc, деградации хитина и/или деградации целлобиозы), клетки по меньшей мере одного вида микроорганизмов с активностью метаболизма яблочной кислоты (типа ассимиляции яблочной кислоты), клетки по меньшей мере одного вида микроорганизмов с активностью продукции фитогормонов (типа индола (ауксина)), клетки по меньшей мере одного вида микроорганизмов с активностью метаболизма железа (типа связывания железа (сидерофоры)) и/или клетки по меньшей мере одного вида микроорганизмов с активностью дефосфорилирования органического фосфата.
Композиция может включать клетки микроорганизмов с одной или несколькими (как-то 2 и более, 3 и более, 4 и более, 5 и более, 6 и более, 7 и более или всеми) из вышеуказанных характеристик или функций, а также и с другими желательными характеристиками или функциями. В определенных примерах микробиологическая композиция включает клетки трех и более видов микроорганизмов, каждый из которых обладает по меньшей мере одной из приведенных функциональностей. В некоторых примерах композиция включает клетки по меньшей мере одного вида микроорганизмов с активностью азотного метаболизма и клетки по меньшей мере одного вида микроорганизмов с устойчивостью к соли; или клетки по меньшей мере одного вида микроорганизмов с активностью азотного метаболизма, клетки по меньшей мере одного вида микроорганизмов с устойчивостью к соли, клетки по меньшей мере одного вида микроорганизмов с активностью солюбилизации солей кальция и/или фосфата и/или цинка и клетки по меньшей мере одного вида микроорганизмов с целлюлолитической/хитинолитической активностью; или клетки по меньшей мере одного вида микроорганизмов с активностью азотного метаболизма, клетки по меньшей мере одного вида микроорганизмов с устойчивостью к соли, клетки по меньшей мере одного вида микроорганизмов с активностью солюбилизации солей кальция и/или фосфата и/или цинка, клетки по меньшей мере одного вида микроорганизмов с целлюлолитической/хитинолитической активностью и клетки по меньшей мере одного вида микроорганизмов с активностью метаболизма яблочной кислоты; или же клетки по меньшей мере одного вида микроорганизмов с активностью азотного метаболизма, клетки по меньшей мере одного вида микроорганизмов с устойчивостью к соли, клетки по меньшей мере одного вида микроорганизмов с активностью солюбилизации солей кальция и/или фосфата и/или цинка, клетки по меньшей мере одного вида микроорганизмов с целлюлолитической/хитинолитической активностью, клетки по меньшей мере одного вида микроорганизмов с активностью метаболизма яблочной кислоты и клетки по меньшей мере одного вида микроорганизмов с активностью продукции фитогормонов; или же клетки по меньшей мере одного вида микроорганизмов с активностью азотного метаболизма, клетки по меньшей мере одного вида микроорганизмов с устойчивостью к соли, клетки по меньшей мере одного вида микроорганизмов с активностью солюбилизации солей кальция и/или фосфата и/или цинка, клетки по меньшей мере одного вида микроорганизмов с целлюлолитической/хитинолитической активностью, клетки по меньшей мере одного вида микроорганизмов с активностью метаболизма яблочной кислоты, клетки по меньшей мере одного вида микроорганизмов с активностью продукции фитогормонов и клетки по меньшей мере одного вида микроорганизмов с активностью метаболизма железа; или же клетки по меньшей мере одного вида микроорганизмов с активностью азотного метаболизма, клетки по меньшей мере одного вида микроорганизмов с устойчивостью к соли, клетки по меньшей мере одного вида микроорганизмов с активностью солюбилизации солей кальция и/или фосфата и/или цинка, клетки по меньшей мере одного вида микроорганизмов с целлюлолитической/хитинолитической активностью, клетки по меньшей мере одного вида микроорганизмов с активностью метаболизма яблочной кислоты, клетки по меньшей мере одного вида микроорганизмов с активностью продукции фитогормонов, клетки по меньшей мере одного вида микроорганизмов с активностью метаболизма железа и клетки по меньшей мере одного вида микроорганизмов с активностью дефосфорилирования органического фосфата. Эти комбинации микроорганизмов с указанными активностями приводятся только для примера, а здесь предусмотрены любые факторные комбинации указанных активностей. Как изложено здесь, один вид микроорганизмов может обладать более чем одной из перечисленных активностей, поэтому в некоторых примерах композиции, включающие клетки с заданным количеством перечисленных активностей, не обязательно будут содержать клетки такого же числа различных видов микроорганизмов.
Типичные методы определения метаболических характеристик или функций клеток микроорганизмов и идентификации видов микроорганизмов с определенными характеристиками описаны в примере 3. Рядовым специалистам должно быть известно, что один вид микроорганизмов может иметь более чем одну из этих характеристик (к примеру, см. табл. 11 ниже). В описанных ниже примерах виды микроорганизмов идентифицируются по названиям; эти идентификации включают такие виды микроорганизмов, у которых последовательности 16S-рДНК по меньшей мере на 95% (как-то по меньшей мере на 96%, 97%, 98%, 99% или даже на 100%) идентичны последовательностям, связанным с каждым из этих поименованных видов и приведенным здесь как SEQ ID NOs: 3-24.
Так, в некоторых воплощениях представленные композиции включают клетки по меньшей мере одного (как-то по меньшей мере 2, по меньшей мере 3, по меньшей мере 4, по меньшей мере 5, по меньшей мере 6, по меньшей мере 7, по меньшей мере 8, по меньшей мере 9, по меньшей мере 10 и более) вида микроорганизмов с активностью азотного метаболизма, к примеру, по меньшей мере одного из Acetobacter pasteurianus, Azotobacter vinelandii, Bacillus megaterium, Bacillus subtilis, Bacillus licheniformis, Oceanobacillus oncorhynchi, Bacillus amyloliquefaciens, Bacillus flexus, Virgibacillus halophilus, Clostridium beijerinckii, Clostridium pasteurianum, Paenibacillus cookii, Paenibacillus lautus (например, близкородственных Paenibacillus lautus и Paenibacillus sp. штамма Y412MC10, к примеру, видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 12), Pseudomonas sp. (близкородственных P. entomophila, P. fluorescens и P. putida, к примеру, видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 17) и Streptomyces griseus. В других примерах представленные композиции включают клетки по меньшей мере одного (как-то по меньшей мере 2, по меньшей мере 3, по меньшей мере 4, по меньшей мере 5, по меньшей мере 6, по меньшей мере 7, по меньшей мере 8, по меньшей мере 9, по меньшей мере 10 и более) вида микроорганизмов с устойчивостью к 5% NaCl, к примеру, Bacillus megaterium, Bacillus subtilis, Bacillus licheniformis, Oceanobacillus oncorhynchi, Bacillus amyloliquefaciens, Bacillus sp. (близкородственных B. kochii, B. pocheonensis и Bacillus sp. штамма R-27341, к примеру, видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 15), Bacillus flexus, Lactobacillus casei/paracasei, Paenibacillus lautus (например, близкородственных Paenibacillus lautus и Paenibacillus sp. штамма Y412MC10, к примеру, видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 12), Pseudomonas putida и Pseudomonas sp. (близкородственных P. entomophila, P. fluorescens и P. putida, к примеру, видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 17). В других примерах представленные композиции включают клетки по меньшей мере одного (как-то по меньшей мере 2, по меньшей мере 3, по меньшей мере 4, по меньшей мере 5, по меньшей мере 6, по меньшей мере 7 и более) вида микроорганизмов с активностью солюбилизации солей фосфата и/или кальция и/или цинка, к примеру, по меньшей мере одного из Clostridium beijerinckii, Clostridium pasteurianum, Lactobacillus casei/paracasei, Lactobacillus buchneri, Lactobacillus vini, Lactobacillus delbrueckii и Paenibacillus lautus (например, близкородственных Paenibacillus lautus и Paenibacillus sp. штамма Y412MC10, к примеру, видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 12). А еще в одних примерах представленные композиции включают клетки по меньшей мере одного (как-то по меньшей мере 2, по меньшей мере 3, по меньшей мере 4, по меньшей мере 5, по меньшей мере 6, по меньшей мере 7, по меньшей мере 8, по меньшей мере 9, по меньшей мере 10 и более) вида микроорганизмов с целлюлолитической и/или хитинолитической активностью, к примеру, одного или нескольких из Bacillus megaterium, Bacillus subtilis, Bacillus amyloliquefaciens, Bacillus sp. (близкородственных B. kochii, B. pocheonensis и Bacillus sp. штамма R-27341, к примеру, видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 15), Bacillus flexus, Clostridium beijerinckii, Clostridium pasteurianum, Lactobacillus casei/paracasei, Lactobacillus vini, Paenibacillus lautus (например, близкородственных Paenibacillus lautus и Paenibacillus sp. штамма Y412MC10, к примеру, видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 12), Paenibacillus chibensis и Streptomyces griseus. В других примерах представленные композиции включают клетки по меньшей мере одного (как-то по меньшей мере 2, по меньшей мере 3, по меньшей мере 4, по меньшей мере 5, по меньшей мере 6 и более) вида микроорганизмов с активностью метаболизма яблочной кислоты, к примеру, по меньшей мере одного из Bacillus megaterium, Bacillus subtilis, Bacillus licheniformis, Bacillus amyloliquefaciens, Bacillus sp. (близкородственных B. kochii, B. pocheonensis и Bacillus sp. штамма R-27341, к примеру, видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 15), Bacillus flexus, Paenibacillus cookii, Paenibacillus lautus (например, близкородственных Paenibacillus lautus и Paenibacillus sp. штамма Y412MC10, к примеру, видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 12), Paenibacillus chibensis, Pseudomonas putida, Pseudomonas sp. (близкородственных P. entomophila, P. fluorescens и P. putida, к примеру, видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 17) и Streptomyces griseus. Еще в одних примерах представленные композиции включают клетки по меньшей мере одного (как-то по меньшей мере 2, по меньшей мере 3 и более) вида микроорганизмов с активностью продукции фитогормонов (например, индола (ауксина)), к примеру, по меньшей мере одного из Clostridium pasteurianum, Lactobacillus vini и Lactobacillus buchneri. А в других примерах представленные композиции включают клетки по меньшей мере одного (как-то по меньшей мере 2, по меньшей мере 3 и более) вида микроорганизмов с активностью метаболизма железа (например, связывания железа), к примеру, по меньшей мере одного из Clostridium pasteurianum и Clostridium beijerinckii. Также предусмотрены композиции, включающие клетки микроорганизмов с любой комбинацией этих характеристик или активностей.
В дополнительных воплощениях представленные здесь микробиологические композиции могут дополнительно включать клетки одного или нескольких (как-то 2 и более, 3 и более, 4 и более или всех) из Desulfosporosinus meridiei, Nitrosopumilus sp., Marinobacter bryozoorum, Leptospirillum ferrodiazotrophum и Lactobacillus acidophilus.
Представленные композиции могут включать в себя один или несколько дополнительных компонентов в дополнение к микроорганизмам, включая, без ограничения, соли, ионы металлов и/или буферы (к примеру, одно или несколько из KH2PO4, K2HPO4, CaCl2, MgSO4, FeCl3, NaMoO4 и/или Na2MoO4), микроэлементы (такие как сера, сульфат, сульфит, медь или селен), микронутриенты (как-то бор (B), цинк (Zn), марганец (Mn), железо (Fe), медь (Cu), молибден (Мо), хлор (Cl)), витамины (как-то витамины B или витамин K), сахара (такие как сахароза, глюкоза или фруктоза), хитин, хитозан, глюкозамин, белок и/или одну или несколько аминокислот. Дополнительные компоненты, которые также могут входить в композиции, включают HYT B, HYT C и/или HYT D, одно или несколько удобрений (например, жидкое удобрение), один или несколько пестицидов, один или несколько фунгицидов, один или несколько гербицидов, один или несколько инсектицидов, один или несколько растительных гормонов, один или несколько растительных элиситоров либо комбинации из двух или нескольких из этих компонентов.
В некоторых воплощениях представленные композиции находятся в жидкой среде (типа культуральной или ферментационной среды или среды хранения) или в инокуляте. В других воплощениях композиции находятся на твердой или студенистой среде (типа культуральных чашек), содержащей или поддерживающей микроорганизмы. В других примерах представленные композиции являются лиофилизованными и могут быть восстановлены путем добавления жидкости (типа культуральной среды) и выращивания в жидкой среде или путем нанесения штрихом на твердую среду.
В других воплощениях описанные здесь композиции присутствуют в сухом виде типа сухого порошка, брикета или гранул. Сухие формы могут быть получены путем добавления осмопротектора (типа сахара, к примеру, трегалозы и/или мальтодекстрина) в микробиологические композиции в виде раствора в требуемом соотношении. Этот раствор объединяют с сухим носителем или абсорбирующим веществом типа древесной муки или глины при желательной концентрации микробиологической композиции (типа 2-30%, к примеру, 2,5-10%, 5-15%, 7,5-20% или 15-30%). Гранулы могут быть получены путем включения глинистых или полимерных связующих, которые служат для удержания гранул вместе или придают определенные физические свойства или свойства разложения. Типичные способы формирования гранул включают ротационную грануляцию, грануляцию в смесителе или экструзию. В других примерах сухие формы получают путем распыления или замачивания описанной здесь жидкой микробиологической композиции на/в твердом носителе типа бентонита или же нанесения жидкой микробиологической композиции непосредственно на гранулы удобрения. В других примерах сухие формы включают композиции, содержащие клетки одного или нескольких описанных здесь видов микроорганизмов (либо их комбинации), которые были подвергнуты лиофилизации (лиофилизованы). Рядовым специалистам в данной области известны и другие способы получения сухих форм, включающих в себя один или несколько видов микроорганизмов, к примеру, как описано в Formulation of Microbial Biopesticides: Beneficial Microorganisms, Nematodes and Seed Treatments. Burges, ed., Springer Science, 1998.
В некоторых примерах композиции содержатся при температуре, способствующей росту микроорганизмов, например, при 25-45°C (как-то при 30-35°C, при 30-40°C или при 35-40°C). В других примерах композиции хранятся при такой температуре, при которой микроорганизмы не растут или являются неактивными, как-то менее 25°C (к примеру, 20°C, 15°C, 10°C, 4°C, -20°C, -40°C, -70°C или меньше). Специалисты в данной области могут составить композиции для хранения в холодильнике, к примеру, путем включения стабилизаторов (типа глицерина). В следующих примерах композиции хранятся при температуре окружающей среды типа 0-35°C (к примеру, при 10-30°C или при 15-25°C).
B Способы получения определенных композиций
В некоторых воплощениях представленные композиции получают путем совместного культивирования или выращивания клеток двух или нескольких приведенных видов микроорганизмов (как-то 2 и более, 3 и более, 4 и более, 5 и более, 6 и более, 7 и более, 8 и более, 9 и более, 10 и более, 11 и более, 12 и более, 13 и более, 14 и более, 15 и более, 16 и более, 17 и более, 18 и более, 19 и более, 20 и более, 21 и более или 22 и более видов микроорганизмов). В тех примерах, где совместно культивируются не все виды микроорганизмов в композиции, композиции получают путем объединения двух или нескольких совместно культивируемых подмножеств (субкомпозиций) различных микроорганизмов в композиции. Дополнительные компоненты (например, немикробные компоненты) могут присутствовать во время получения смеси различных микроорганизмов (полного набора или подмножества) или же могут быть добавлены после получения смеси различных микроорганизмов в композиции.
В некоторых примерах представленные композиции получают путем совместного культивирования всех видов микроорганизмов в композиции. Так, в одном примере представленные композиции получают при совместном культивировании клеток каждого из видов Lactobacillus delbrueckii, Virgibacillus halophilus, Azotobacter vinelandii, Clostridium pasteurianum, Paenibacillus chibensis, Streptomyces griseus, Pseudomonas sp. (близкородственных P. entomophila, P. fluorescens и P. putida, к примеру, видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 17), Pseudomonas putida, Bacillus sp. (близкородственных B. kochii, B. pocheonensis и Bacillus sp. штамма R-27341, к примеру, видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 15), Bacillus amyloliquefaciens, Oceanobacillus oncorhynchi, Paenibacillus lautus, Bacillus licheniformis, Lactobacillus vini, Paenibacillus cookii, Bacillus subtilis, Lactobacillus buchneri, Bacillus megaterium, Acetobacter pasteurianus, Clostridium beijerinckii, Lactobacillus casei/paracasei и Bacillus flexus. В другом примере представленные композиции получают при совместном культивировании клеток каждого из видов Lactobacillus delbrueckii, Virgibacillus halophilus, Azotobacter vinelandii, Clostridium pasteurianum, Streptomyces griseus, Pseudomonas sp. (близкородственных P. entomophila, P. fluorescens и P. putida, к примеру, видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 17), Pseudomonas putida, Bacillus sp. (близкородственных B. kochii, B. pocheonensis и Bacillus sp. штамма R-27341, к примеру, видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 15), Bacillus amyloliquefaciens, Oceanobacillus oncorhynchi, Bacillus licheniformis, Lactobacillus vini, Bacillus subtilis, Lactobacillus buchneri, Bacillus megaterium, Acetobacter pasteurianus, Clostridium beijerinckii, Lactobacillus casei/paracasei и Bacillus flexus. Еще в других примерах представленные композиции получают при совместном культивировании клеток каждого из видов Lactobacillus delbrueckii, Virgibacillus halophilus, Azotobacter vinelandii, Clostridium pasteurianum, Paenibacillus chibensis, Streptomyces griseus, Pseudomonas sp. (близкородственных P. entomophila, P. fluorescens и P. putida, к примеру, видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 17), Pseudomonas putida, Bacillus sp. (близкородственных B. kochii, B. pocheonensis и Bacillus sp. штамма R-27341, к примеру, видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 15), Bacillus amyloliquefaciens, Oceanobacillus oncorhynchi, Paenibacillus lautus, Bacillus licheniformis, Lactobacillus vini, Paenibacillus cookii, Lactobacillus buchneri, Bacillus megaterium, Acetobacter pasteurianus, Clostridium beijerinckii, Lactobacillus casei/paracasei и Bacillus flexus. В следующем примере представленные композиции получают при совместном культивировании клеток каждого из видов Lactobacillus delbrueckii, Virgibacillus halophilus, Azotobacter vinelandii, Clostridium pasteurianum, Streptomyces griseus, Pseudomonas sp. (близкородственных P. entomophila, P. fluorescens и P. putida, к примеру, видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 17), Pseudomonas putida, Bacillus sp. (близкородственных B. kochii, B. pocheonensis и Bacillus sp. штамма R-27341, к примеру, видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 15), Bacillus amyloliquefaciens, Oceanobacillus oncorhynchi, Bacillus licheniformis, Lactobacillus vini, Lactobacillus buchneri, Bacillus megaterium, Acetobacter pasteurianus, Clostridium beijerinckii, Lactobacillus casei/paracasei и Bacillus flexus.
В другом примере представленные композиции получают при совместном культивировании клеток каждого из видов Lactobacillus delbrueckii, Azotobacter vinelandii, Clostridium pasteurianum, Streptomyces griseus, Pseudomonas sp., Pseudomonas putida, Bacillus amyloliquefaciens, Bacillus licheniformis, Lactobacillus vini, Bacillus subtilis, Lactobacillus buchneri, Bacillus megaterium, Acetobacter pasteurianus, Clostridium beijerinckii, Lactobacillus casei/paracasei и Bacillus flexus. А еще в одних примерах представленные композиции получают при совместном культивировании клеток каждого из видов Lactobacillus delbrueckii, Azotobacter vinelandii, Clostridium pasteurianum, Paenibacillus chibensis, Streptomyces griseus, Pseudomonas sp., Pseudomonas putida, Bacillus amyloliquefaciens, Oceanobacillus oncorhynchi, Paenibacillus lautus, Bacillus licheniformis, Lactobacillus vini, Paenibacillus cookii, Bacillus subtilis, Lactobacillus buchneri, Bacillus megaterium, Acetobacter pasteurianus, Clostridium beijerinckii, Lactobacillus casei/paracasei, и Bacillus flexus.
Культуральные среды, которые можно использовать для получения этих композиций при совместном культивировании, описаны в Примере 2 и включают, без ограничения, среду, содержащую 2% патоки (вес/об.), фосфатно-солевой буфер (PBS), 0,1% белков молочной сыворотки (вес/об.) и 0,25% Ferti-Nitro Plus (вес/об.). В других примерах среда для совместного культивирования микробных клеток включает фосфатно-солевой буфер (1х), черную патоку (2-10% вес/об.), белки молочной сыворотки (0,1-0,5% вес/об.), Ferti-Nitro Plus Plant N (0,25-1,25% вес/об.) с экстрактом ламинарии (0,0067%) или без него, дрожжевой порошок (0,0033% вес/об.) и/или спирулины (0,0067% вес/об.).
В других примерах представленные композиции получают путем совместного культивирования клеток из по меньшей мере двух подгрупп микроорганизмов, а затем объединения этих двух культур с получением композиции. В одном примере композиции получают при совместном культивировании клеток Lactobacillus delbrueckii, Virgibacillus halophilus, Azotobacter vinelandii, Clostridium pasteurianum, Paenibacillus chibensis, Pseudomonas sp. (близкородственных P. entomophila, P. fluorescens и P. putida, к примеру, видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 17), Pseudomonas putida, Bacillus sp. (близкородственных B. kochii, B. pocheonensis и Bacillus sp. штамма R-27341, к примеру, видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 15), Bacillus amyloliquefaciens, Oceanobacillus oncorhynchi, Paenibacillus lautus, Bacillus licheniformis, Lactobacillus vini, Paenibacillus cookii, Bacillus subtilis, Lactobacillus buchneri, Bacillus megaterium, Acetobacter pasteurianus и Lactobacillus casei/paracasei (группа из 19 видов микроорганизмов) и отдельно при совместном культивировании Clostridium beijerinckii, Streptomyces griseus и Bacillus flexus (группа из 3 видов микроорганизмов).
В другом примере композиции получают при совместном культивировании Lactobacillus delbrueckii, Virgibacillus halophilus, Azotobacter vinelandii, Clostridium pasteurianum, Pseudomonas sp. (близкородственных P. entomophila, P. fluorescens и P. putida, к примеру, видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 17), Pseudomonas putida, Bacillus sp. (близкородственных B. kochii, B. pocheonensis и Bacillus sp. штамма R-27341, к примеру, видов микроорганизмов, у которых последовательность 16S-рРНК по меньшей мере на 99% идентична последовательности SEQ ID NO: 15), Bacillus amyloliquefaciens, Oceanobacillus oncorhynchi, Bacillus licheniformis, Lactobacillus vini, Bacillus subtilis, Lactobacillus buchneri, Bacillus megaterium, Acetobacter pasteurianus и Lactobacillus casei/paracasei (группа из 16 видов микроорганизмов) и отдельно при совместном культивировании Clostridium beijerinckii, Streptomyces griseus и Bacillus flexus. После индивидуального совместного культивирования двух смесей в условиях, достаточных для роста микроорганизмов (типа описанных в примере 2), индивидуальные совместные культуры смешивают, получая композицию.
В некоторых примерах среда, используемая для совместного культивирования группы из 19 видов микроорганизмов или группы из 16 видов микроорганизмов, включает 10% патоки (вес/об.), 1x PBS, 0,5% белков молочной сыворотки (вес/об.) и 1,25% Ferti-Nitro Plus (вес/об.). В некоторых примерах среда, используемая для совместного культивирования группы из 3 видов микроорганизмов, включает 2% патоки (вес/об.), 1x PBS, 0,1% белков молочной сыворотки (вес/об.) и 0,25% Ferti-Nitro Plus. Однако рядовые специалисты в данной области могут определить и другие среды, которые подходят для совместного культивирования микроорганизмов, содержащие различные количества перечисленных ингредиентов, как описано в примере 2. После индивидуального совместного культивирования двух смесей в условиях, достаточных для роста микроорганизмов (типа описанных в примере 2), индивидуальные совместные культуры (типа группы из 19 микроорганизмов и группы из 3 микроорганизмов или группы из 16 микроорганизмов и группы из 3 микроорганизмов) смешивают, получая композицию. В некоторых примерах две совместные культуры смешивают при соотношении большой группы (группы из 19 видов микроорганизмов или группы из 16 видов микроорганизмов) и малой группы (группы из 3 видов микроорганизмов) от 10:1 до 1:0,5 (как-то от 8:1 до 1:1, от 5:1 до 2:1, от 3:1 до 1:0,5). В некоторых примерах соотношение совместных культур составляет 10:1, 9:1, 8:1, 7:1, 6:1, 5:1, 4:1, 3:1, 2:1, 1:1 или 1:0,5. В предпочтительных воплощениях соотношение совместных культур составляет 6,5:1, 1:1, 2:1 или 1:0,5. Аналогичные среды для совместного культивирования и смеси могут использоваться с такими культурами, которые идентичны перечисленным выше, но не включают Bacillus subtilis.
В других примерах композиции получают путем раздельного культивирования микроорганизмов под депозитными номерами PTA-123288 и PTA-123289 в ATCC и смешивания индивидуальных культур с получением композиции. Еще в других примерах композиции получают путем раздельного культивирования микроорганизмов под депозитными номерами PTA-123298 и PTA-123289 в ATCC и смешивания индивидуальных культур с получением композиции.
III Процессы биодеградации
Представленные композиции могут применяться для разложения биологических материалов типа богатых хитином материалов, к примеру, водных животных или побочных продуктов из водных животных, насекомых или грибов. Так, в некоторых воплощениях изложены способы, включающие смешивание одной или нескольких из представленных микробиологических композиций с содержащим хитин биологическим материалом с образованием смеси и ферментацию смеси. В некоторых воплощениях способы также включают разделение смеси на твердую, водную и необязательно липидную фракцию (фиг. 1).
В некоторых воплощениях изложенный здесь процесс биодеградации включает смешивание представленной здесь микробиологической композиции с одним или несколькими содержащими хитин биологическими материалами. Содержащие хитин биологические материалы включают, без ограничения, водных животных или побочные продукты из водных животных, насекомых и грибов. В некоторых примерах содержащий хитин биологический материал представляет собой водное животное типа водных членистоногих (к примеру, представителей класса Malacostraca). Водные членистоногие для применения в изложенных способах включают креветок, крабов, омаров, раков и криль. В некоторых примерах в изложенных здесь способах биодеградации используются целые водные животные (типа водных членистоногих) или побочные продукты из водных животных. Побочные продукты водных животных включают любые части водных животных, как-то любые части, полученные при переработке водных животных. В некоторых примерах побочными продуктами водных животных являются целые экзоскелеты или части экзоскелетов водных животных типа панцирей креветок, крабов, раков или омаров. В других примерах побочными продуктами водных животных являются части водных животных, к примеру, цефалоторакс креветок.
В других примерах хитин-содержащий биологический материал включает грибы типа грибов из филума Zygomycota, Basidiomycota, Ascomycota или Deuteromycota. Конкретные типичные грибы включают Aspergillus spp., Penicillium spp., Trichoderma spp., Saccharomyces spp. и Schizosaccharomyces spp. Таким образом, источником содержащего хитин биологического материала могут служить отходы пекарного, пивоваренного и спиртоводочного производства. В других примерах хитин-содержащий биологический материал включает насекомых, содержащих хитин в своих экзоскелетах, типа кузнечиков, сверчков, жуков и других насекомых. Также предусматривается, что источниками хитина могут служить побочные продукты переработки таких насекомых.
Хитин-содержащий биологический материал смешивают с композицией, описанной выше в разделе II, до получения практически однородной смеси. В некоторых примерах хитин-содержащий биологический материал измельчают, дробят, крошат, размалывают или иным образом диспергируют перед смешиванием с представленной здесь микробиологической композицией. В конкретных примерах смесь содержит 10-50% (как-то 10-20%, 20-30%, 30-40%, 25-40%, к примеру, около 25%, около 30%, около 35%, около 40%, около 45% или около 50%) хитин-содержащего материала (типа головы креветок) (вес/об.) в инокуляте, содержащем 0,1-5% (как-то 0,1-1%, 0,5-2%, 1-2%, 2-3%, примерно 0,1%, 0,2%, 0,3%, 0,5%, 0,8%, 1%, 1,25%, 1,5%, 1,75%, 2%, 2,5%, 3%, 4% или 5%) микробиологической композиции (об./об.).
В некоторых примерах смешивают вместе инокулят, содержащий хитин биологический материал и сахар (или другой источник углерода), к примеру, путем перемешивания или встряхивания. В других примерах один или несколько микроорганизмов в микробиологической композиции необязательно подвергают активации перед смешиванием с хитин-содержащим биологическим материалом и ферментацией. Для изложенных здесь способов активация не требуется. Специалисты в данной области могут корректировать время и/или температуру при ферментации в зависимости от того, проводится ли активация микроорганизмов перед ферментацией. Активация микроорганизмов может проводиться путем инкубации инокулята микробиологической композиции с источником углерода (типа сахара, к примеру, с глюкозой, сахарозой, фруктозой или другим сахаром) при температуре и в течение достаточного времени для роста микроорганизмов. В некоторых примерах концентрация инокулята микроорганизмов (типа описанной здесь микробиологической композиции) составляет 0,05-5% об./об. (к примеру, 0,5-5%, 0,5-2%, 1-2% или 2-3%) в жидкой среде. Инокулят разводят в растворе, содержащем 0,1-1% сахара (к примеру, 0,1-0,5%, 0,1-0,3%, 0,2-0,6% или 0,5-1%, как-то примерно 0,1%, 0,2%, 0,3%, 0,4%, 0,5%, 0,6%, 0,7%, 0,8%, 0,9% или 1%) и инкубируют при температуре окружающей среды, к примеру, при 20-40°C (как-то при 20°C, при 25°C, при 30°C, при 35°C или при 40°C) в течение 1-5 дней (как-то примерно 24 часов, 48 часов, 72 часов, 96 часов или 120 часов). В других примерах активация микроорганизмов может проводиться путем инкубации инокулята микробиологической композиции при температуре и в течение достаточного времени для роста микроорганизмов, к примеру, путем инкубации при 20-40°C (как-то 25-35°C) в течение от 12 часов до 5 дней (как-то 1-4 дней или 2-3 дней). В некоторых неограничительных примерах микроорганизмы считаются активированными, когда культура достигнет оптической плотности > 0,005 при 600 нм.
После смешивания хитинсодержащего биологического материала и микробиологической композиции (которая необязательно активирована) смесь подвергается ферментации. В некоторых примерах перед ферментацией измеряют pH смеси. При необходимости перед ферментацией доводят значение pH до выбранного диапазона (например, рН от 3 до 4 или от 3,5 до 4). Смесь инкубируют при температуре 20-40°C (к примеру, при 30-36°C, как-то при 30°C, при 31°C, при 32°C, при 33°C, при 34°C, при 35°C, при 36°C, при 37°C, при 38°C, при 39°C или при 40°C) в течение 1-30 дней (как-то 3-28 дней, 7-21 дня, примерно 3, 5, 7, 10, 14, 16, 20, 24, 28 или 30 дней). Смесь периодически перемешивают (к примеру, при непрерывном встряхивании). В некоторых примерах смесь встряхивают на протяжении 1-7 дней, к примеру, через каждые 1, 2, 3, 4, 5, 6 или 7 дней. В некоторых неограничительных примерах ферментация продолжается до тех пор, пока титруемая кислотность (TTA) не составит 3-5%, а pH не составит 4-5.
После ферментации полученную ферментированную смесь разделяют по меньшей мере на твердую и жидкую фракции. В некоторых примерах твердая фракция именуется “HYT C”, а в некоторых примерах жидкая фракция именуется “HYT B”. В некоторых примерах ферментация переносится из резервуара в емкость для осаждения. После этого жидкость декантируют и центрифугируют. В одном неограничительном примере ферментированную смесь центрифугируют при 1250 об/мин (930×g) в течение 15 мин при 5°C, получая жидкую и липидную (например, пигментную) фракцию. Жидкая (или водная) фракция, полученная в процессе биодеградации, может храниться при температуре окружающей среды. В некоторых неограничительных примерах в жидкую фракцию добавляют сахар, к примеру, 1-10% по объему.
Жидкая фракция может включать такие компоненты, как белок, аминокислоты, глюкозамин, микроэлементы (как-то кальций, магний, цинк, медь, железо и/или марганец) и/или ферменты (как-то молочные ферменты, протеазы, липазы и/или хитиназы). В некоторых неограничительных примерах жидкая фракция содержит (вес/об.) 1-5% общих аминокислот, 3-7% белка, 0,1-2% азота, менее 0,2% фосфора, 0,5-1% калия, 4-8% углерода, 0,2-1% кальция, менее 0,2% магния, менее 0,2% натрия и/или 0,1-0,4% серы. В дополнительных неограничительных примерах жидкая фракция содержит 0,01-0,2% глюкозамина (к примеру, 0,1% или меньше). Жидкая фракция также может содержать один или несколько микроорганизмов (например, из инокулята, использовавшегося для запуска процесса ферментации) и/или следовые количества хитозана или хитина. Жидкая фракция в некоторых примерах именуется как “HYT B”.
Твердая фракция, полученная в процессе биодеградации, содержит хитин (к примеру, 50-70% или 50-60% хитина). Твердая фракция также может содержать один или несколько микроэлементов (как-то кальций, магний, цинк, медь, железо и/или марганец), белок или аминокислоты и/или один или несколько микроорганизмов из инокулята, использовавшегося для запуска процесса ферментации. Твердая фракцию в некоторых примерах именуется как “HYT C”. HYT C необязательно подвергают микронизации с получением микронизованного и остаточного хитина. В некоторых неограничительных примерах твердая фракция содержит (вес/об.) 9-35% общих аминокислот, 30-50% сырого белка, 5-10% азота, 0,3-1% фосфора, меньше 0,3% калия, 35-55% углерода, 0,5-2% кальция, менее 0,1% магния, 0,1-0,4% натрия и/или 0,2-0,5% серы.
В некоторых примерах также отделяют липидную фракцию от твердой и жидкой фракций. Липидная фракция является верхней фазой жидкой фракции. Липидная фракция содержит такие соединения, как стеролы, витамин A и/или витамин E, жирные кислоты (типа DHA и/или EHA), а в некоторых примерах и каротиноидные пигменты (к примеру, астаксантин). Липидная фракция может применяться для различных целей, включая, без ограничения, производство косметических или пищевых продуктов.
В дополнительных воплощениях хитин подвергается ферментации с помощью представленной микробиологической композиции. В некоторых примерах хитин (типа HYT C или микронизированный и/или остаточный хитин, полученный, как описано выше) смешивают с микробиологической консорцией или композицией, содержащей описанные здесь микроорганизмы и гидролизат белка (например, HYT B), и проводят ферментацию до получения ферментированной смеси. В результате ферментации по меньшей мере часть хитина в исходной смеси расщепляется. В некоторых примерах смесь инкубируют при температуре 20-40°C (к примеру, при 30-35°C, как-то при 30°C, при 31°C, при 32°C, при 33°C, при 34°C, при 35°C, при 36°C, при 37°C, при 38°C, при 39°C или при 40°C) в течение от 1 дня до 30 дней (как-то 2-28 дней, 4-24 дней, 16-30 дней, 10-20 дней или 12-24 дней). В некоторых примерах смесь периодически перемешивают (к примеру, при периодическом перемешивании). В других примерах смесь перемешивают непрерывно. В одном неограничительном примере смесь перемешивают в течение 1-12 часов в день (как-то 2-8 часов или 4-10 часов). Можно периодически отслеживать pH ферментационной смеси. В некоторых примерах значение рН необязательно поддерживается на уровне 4-5. В некоторых примерах ферментация продолжается до тех пор, пока общая титруемая кислотность (TTA) не составит по меньшей мере 1-10% (как-то 2-8%, 4-8% или 5-10%).
После ферментации полученную ферментированную смесь разделяют по меньшей мере на твердую и жидкую фракцию, к примеру, путем декантации, фильтрования и/или центрифугирования. Жидкая фракция, получающаяся при ферментации HYT B и хитина с помощью микробиологической композиции, в некоторых примерах упоминается как “HYT D”. В некоторых неограничительных примерах жидкая фракция содержит (вес/об.) 0,5-2% общих аминокислот, 3-7% белка, 0,5-1% азота, менее 0,1% фосфора, 0,4-1% калия, 3-7% углерода, менее 0,5% кальция, менее 0,1% магния, менее 0,3% натрия и/или менее 0,3% серы. Кроме того, HYT D содержит менее 50% хитина (как-то менее 45%, менее 40%, менее 35% или менее 30% хитина) и менее 2% глюкозамина (как-то менее 1,5% или менее 1% глюкозамина). В других примерах HYT D содержит 25-50% хитина и 0,5-2% глюкозамина.
IV Способы обработки почвы, растений и/или семян
Представленные микробиологические композиции, по отдельности или в сочетании с представленными здесь продуктами (типа HYT B, HYT C и/или HYT D), могут применяться для обработки почвы, растений или частей растения (как-то корней, стеблей, листвы, семян или побегов). Способы получения HYT B, HYT C и HYT D описаны в разделе III (выше), а также в U.S. Pat. No. 8,748,124 и International Pat. App. Publ. No. WO 2012/175738, которые оба включены сюда путем ссылки во всей полноте.
В некоторых примерах обработка представленными композициями улучшает рост растений, улучшает стрессоустойчивость и/или повышает урожайность. В некоторых воплощениях способы включают контактирование почвы, растений (как-то листвы растений, стеблей, корней, побегов или других частей растений) или семян с представленными здесь микробиологическими композициями. Способы также могут включать выращивание обработанных растений, частей растений или семян и/или культивирование растений, частей растений или семян в обработанной почве.
Микроорганизмы в композиции необязательно подвергают активации перед применением. В некоторых примерах активация микроорганизмов проводится так же, как описано выше в разделе III. В других примерах микроорганизмы активируются путем смешивания 100 долей воды и 1 доли микробиологической композиции и инкубации при 15-40°C (как-то при 20-40°C, при 15-30°C или при 25-35°С) в течение от 12 часов до 14 дней (как-то 1-14 дней, 3-10 дней, 3-5 дней или 5-7 дней). Смесь для активации необязательно может также включать 1 часть HYT B, если микробиологическая композиция будет применяться в сочетании с HYT B.
В других воплощениях способы включают контактирование почвы, растений, частей растений или семян с представленной композицией и одним или несколькими из HYT B, HYT C и HYT D (как-то с одним, двумя или всеми из HYT B, HYT C и HYT D). HYT B, HYT C и/или HYT D можно наносить на почву, растения (или части растений) и/или семена по отдельности, к примеру, последовательно, одновременно или практически одновременно с представленными микробными композициями.
В некоторых примерах способы включают контактирование почвы, растений (или частей растений) или семян с представленной микробиологической композицией и одним или несколькими дополнительными компонентами, включая, без ограничения, хитин, хитозан, глюкозамин, белок, аминокислоты, жидкие удобрения, один или несколько пестицидов, один или несколько фунгицидов, один или несколько гербицидов, один или несколько инсектицидов, один или несколько растительных гормонов, один или несколько растительных элиситоров либо комбинации из двух или нескольких из них. Дополнительные компоненты могут входить в композицию, содержащую приведенные здесь микроорганизмы, или же их можно наносить на почву, растения (или части растений) и/или семена отдельно, к примеру, последовательно, одновременно или практически одновременно с представленными композициями.
В определенных воплощениях микробиологическая композиция объединяется с жидким удобрением (к примеру, водным раствором или суспензией, содержащей растворимый азот). В некоторых примерах жидкое удобрение включает органический источник азота типа мочевины либо азотсодержащую неорганическую соль типа гидроксида аммония, нитрата аммония, сульфата аммония, пирофосфата аммония, тиосульфата аммония либо их комбинации. В качестве растворимого азота также можно использовать жидкий аммиак (20-24,6% безводного аммиака). В некоторых примерах микробиологическая консорция или композиция объединяется с жидким удобрением (к примеру, смешивается с жидким удобрением) непосредственно перед применением или за короткое время до применения (типа в пределах от 10 минут до 24 часов перед применением, к примеру, за 30 минут, 1 час, 2 часа, 3 часа, 4 часа, 6 часов, 8 часов, 12 часов, 16 часов, 18 часов или 24 часа до применения). В других примерах микробиологическая консорция или композиция объединяется с жидким удобрением (к примеру, смешивается с жидким удобрением) по меньшей мере за 24 часа до применения (как-то от 24 часов до 6 месяцев, к примеру, по меньшей мере за 36 часов, за 48 часов, за 72 часа, за 96 часов, за 1 неделю, за 2 недели, за 4 недели, за 8 недель или за 12 недель до применения).
В некоторых примерах рассчитывается наносимое количество композиции (например, на 1 акр или гектар) и композиция разбавляется водой (или в некоторых примерах жидким удобрением) до количества, достаточного для опрыскивания или орошения обрабатываемого участка (если композиция жидкая). Композиции можно применять при внесении семян из расчета 0,5-2 литра на акр (как-то 0,5 л/акр, 1 л/акр, 1,5 л/акр или 2 л/акр). Микробиологические композиции также можно наносить на почву (например, возле корней растений) или на растения один или несколько раз во время роста, в таком же или другом количестве. В других примерах композиции можно смешивать с разбавленными гербицидами, инсектицидами, пестицидами или химикатами, регулирующими рост растений. Если наносимая композиция является твердой (типа сухого состава), то её можно наносить прямо на почву, растения или части растений или же суспендировать либо растворить в воде (или другой жидкости) перед применением.
Представленные микробиологические композиции (по отдельности или в сочетании с другими приведенными здесь компонентами) можно вносить различными способами на разных стадиях развития растений, в зависимости от ситуации с культурами и практики ведения сельского хозяйства. В некоторых примерах представленные микробиологические композиции смешивают с жидким удобрением и наносят при внесении семян из расчета 0,5-2 литра на акр (как-то 0,5 л/акр, 1 л/акр, 1,5 л/акр или 2 л/акр) или же наносят индивидуально. Микробиологические композиции и жидкие удобрения также можно наносить на почву (например, возле корней растений) или на растения один или несколько раз во время роста, в таком же или другом количестве. В других примерах представленные микробиологические композиции смешивают с HYT B, разбавляют и наносят при внесении семян из расчета 0,5-2 литра на акр (как-то 0,5 л/акр, 1 л/акр, 1,5 л/акр или 2 л/акр) или же наносят индивидуально. Микробиологические композиции и HYT B также можно наносить на почву (например, возле корней растений) или на растения один или несколько раз во время роста, в таком же или другом количестве.
Еще в других примерах представленные микробиологические композиции и дополнительные компоненты (как-то жидкие удобрения, HYT B или другие компоненты) разбавляют и вносят вместе посредством капельного орошения в низкой концентрации по мере появления побегов или саженцев, вносят при поливе или распыляют в виде разбавленной смеси с питательными веществами при дождевании или капельном орошении в теплицах на побеги или укоренившиеся растения или же вносят индивидуально. А еще в других примерах представленные микробиологические композиции добавляют к другим обработкам почвы в полевых условиях типа добавки при обработке инсектицидами, для удобства. В других примерах, как-то в теплицах, представленные микробиологические композиции и HYT B применяются по отдельности или вместе, в сочетании с жидкими удобрениями (типа рыбных удобрений) и другими питательными веществами, и вводятся через системы дождевания с разбрызгиванием воды или линии капельного орошения на всем протяжении роста растений. В одном примере с теплицами представленные микробиологические композиции и HYT B применяются вместе, к примеру, их разбавляют и вносят при дождевании или внесении удобрений в дозе от 0,25 до 1 л при появлении ростков, а затем от 0,25 до 1 л в середине цикла роста при внесении удобрений и наконец от 0,25 до 1 л при внесении удобрений 5-10 дней в конце цикла роста.
В некоторых воплощениях представленные микробиологические композиции и HYT B применяются вместе или по отдельности (например, последовательно) для повышения урожайности, мощности, зрелости, качества, развития корней или стрессоустойчивости у сельскохозяйственных культур.
Во всех культурах можно вносить в почву HYT C из расчета 0,5-2 кг/акр (как-то 0,5 кг/акр, 1 кг/акр, 1,5 кг/акр или 2 кг/акр) во время появления ростков или посадки, к примеру, в сочетании с представленными микробиологическими композициями. В других примерах HYT C добавляют в растворы для капельного орошения с представленными микробиологическими композициями, а HYT B добавляют в составы для внесения удобрений, содержащие данные микробиологические композиции и HYT B в теплицах, как в приведенных выше примерах.
В дополнительных воплощениях применяется HYT D (по отдельности или в сочетании с представленными здесь микробиологическими композициями или другими компонентами) из расчета 1-20 л/га (как-то 1-15 л/га, 3-10 л/га или 3-5 л/га). В других примерах HYT D (по отдельности или в сочетании с представленными здесь микробиологическими композициями или другими компонентами) применяется для обработки семян для повышения урожайности и производительности культур (к примеру, 1-10 л/кг семян, как-то 1-3 л/кг, 3-5 л/кг или 5-10 л/кг). С другой стороны, HYT D можно вносить в почву (по отдельности или в сочетании с представленными здесь микробиологическими композициями или другими компонентами) из расчета 1-3 л/га для усиления роста растений, к примеру, чтобы растения оставались продуктивными в условиях стресса.
В некоторых примерах обработка почвы, семян, растений или частей растений представленными композициями усиливает рост растений (как-то общий размер растений, количество листвы, количество корней, диаметр корней, длину корней, производство отростков, производство плодов, производство пыльцы и/или производство семян) по меньшей мере на 5% (к примеру, по меньшей мере на 10%, на 30%, на 50%, на 75%, на 100%, по меньшей мере в 2 раза, в 3 раза, в 5 раз, в 10 раз и более). В других примерах представленные способы приводят к повышению урожайности культур на 10-75% (как-то на 20-60% или на 30-50%) по сравнению с необработанными культурами. Другие показатели качества культур включают качество плодов, урожайность, содержание крахмала или сухих веществ, содержание сахара или по шкале Брикса, срок годности плодов или собираемого продукта, производство товарного урожая или заданного размера, качество плодов или продукта, кущение травы и устойчивость к хождению у газонов, опыление и набор плодов, цветение, количество цветков, продолжительность жизни цветков, качество цветения, укореняемость и корневая масса, устойчивость к полеганию, абиотическая стрессоустойчивость к жаре, засухе, холоду и восстановление после стресса, приспособляемость к плохим почвам, уровень фотосинтеза и озеленения и здоровье растений. Для определения эффективности продукции контроли включают такие же агрономические методы без добавления микроорганизмов, выполняемые параллельно.
Представленные способы и композиции могут применяться в отношении любых культур (к примеру, для прямой обработки культур или для обработки почвы до или после посадки). Типичными культурами являются, без ограничения, люцерна, миндаль, бананы, ячмень, брокколи, капуста, канола, морковь, цитрусовые и фруктовые деревья, кукуруза, хлопок, огурцы, цветы и декоративные растения, чеснок, виноград, хмель, садовые растения, лук-порей, дыни, масличные пальмы, лук, арахис и бобовые, ананасы, тополи, сосна и древесные деревья, картофель, малина, рис, кунжут, сорго, соя, кабачки, клубника, сахарный тростник, подсолнечник, помидоры, дерновые и кормовые травы, арбузы, пшеница и эвкалипт.
Примеры
Следующие примеры приводятся для иллюстрации некоторых конкретных особенностей и/или воплощений. Эти примеры не следует рассматривать как ограничение изобретения определенными признаками или описанными воплощениями.
Пример 1. Выделение и идентификация микроорганизмов
В этом примере описаны выделение, идентификация и характеристика микроорганизмов.
Выделение микроорганизмов. Образцы HYT A (Agrinos AS; от 50 мл до 5 л) хранили при комнатной температуре вдали от света. Перед отбором проб из выбранной партии HYT A соответствующий контейнер энергично перемешивали, чтобы обеспечить равномерное распределение содержимого, так как со временем обычно выпадает осадок. Как правило, для выделения микробов оставляли аликвоту в 1-10 мл. В случае Azotobacter проводили выделение из образцов почвы (полученных в N 38° 32′ 49,55″, W 121° 44′ 13,54″).
Выделение на чашках. Из оставшихся аликвот HYT A в асептических условиях отбирали 0,1 мл и смешивали с 9,9 мл стерильной воды или воды с пептоном в культуральной пробирке (разведение 10−2). Затем пробирки перемешивали на вибромешалке (например, 60 секунд при 2000 об/мин) и готовили 10-кратные серийные разведения в воде или воде с пептоном (вплоть до 1:109). После этого по 100 мкл каждого разведения размазывали по полутвердой среде в чашках Петри на 100 мм с помощью стерильного L-образного шпателя. Использовали чашки, содержащие агар для стандартных методов (SMA; BD № 247940), питательный агар (NA; BD № 213000) или другую выбранную питательную среду (таблица 1). Затем инокулированные чашки инкубировали в камерах с контролируемой температурой от 22°C до 35°C. Для выделения анаэробных микробов чашки сначала помещали в анаэробные боксы (например, BD GasPak™ EZ Container Systems, BD Diagnostics), а затем инкубировали при нужной температуре.
Таблица 1. Полутвердые среды, используемые для выделения микроорганизмов из HYT A
* NA: питательный агар (BD # 213000); SMA: агар для стандартных методов (BD # 247940); YPD: дрожжевой экстракт-пептон-декстроза (BD # 242720); АМА: агар со средой для Azotobacter (HIMEDIA # M372); AMAG: агар со средой для Azotobacter с добавлением 10 г/л глюкозы (HIMEDIA # M371); RCM: усиленный агар для Clostridium (BD # 218081); RMA: агар со средой для Rhizobium (HIMEDIA # M408); PA: среда Пиковской (HIMEDIA # M520); MRS: MRS для Lactobacilli (BD # 288210).
Выделение микроорганизмов из образцов почвы. К 30-50 г почвы добавляли 1% масс. маннита или сахарозы, а затем 2 мл стерильной воды на каждые 10 г почвы. Полученный шлам размешивали в стерильной ступке до образования однородной пасты. Затем пасту переносили в чашку Петри и инкубировали при 27-30°C в увлажненной камере вплоть до 1 недели. После этого образец слизистого вещества, образовавшегося на поверхности почвенной пасты, переносили в свежую и стерильную полутвердую среду без азота (см. пример 3 насчет состава среды). Полученный рост микроорганизмов делали биологически чистым путем субкультивирования, как описано ниже.
Получение биологически чистых изолятов. После инкубации чашки подвергали анализу на рост микроорганизмов. Отбирали штаммы микроорганизмов для дальнейшего исследования на основании традиционных макроскопических и микроскопических характеристик колоний, растущих на полутвердых средах (Bergey’s Manual of Systematics of Archaea and Bacteria; Ed., William B. Whitman). Рассматривали такие критерии, как цвет, плотность или морфология (например, форма, высота и края) колоний. Для получения хорошо отделенных колоний на твердой среде использовали метод “штрихования”. Вкратце, отобранные клоны микробов наносили на новую свежую твердую среду и культивировали до появления хорошо дифференцированных колоний. При необходимости проводили несколько пересевов до получения биологически чистых клонов микроорганизмов, которые именуются “изолятами”. Затем на изолятах проводили дополнительные тесты для изучения морфологии бактериальных клеток. Для первоначальной классификации изолятов до соответствующего наиболее вероятного рода также использовали дифференциальное окрашивание по Граму.
Экстракция геномной ДНК микроорганизмов. Выращивали бактериальные клетки различных видов и собирали их из жидкого бульона при оптимизированных условиях культивирования. Для получения геномной ДНК в небольшом объеме использовали набор для выделения ДНК PowerSoil (MoBio, кат. № 12888). Для экстракции геномной ДНК в большом объеме готовили клеточный лизат с помощью набора GenElute Bacterial Genomic DNA (Sigma, кат. № NA2110) или набор буферов для геномной ДНК Qiagen и Genomic-tip 500/G (Qiagen, кат. № 19060 и 10262) в соответствии с рекомендациями производителей. Затем геномную ДНК осаждали равным объемом изопропанола, промывали 70% этанолом, сушили на воздухе и ресуспендировали в буфере TE.
Амплификация и секвенирование генов 16S для проверки таксономии. Амплифицировали полноразмерные гены 16S из различных видов бактерий, используя геномную ДНК и/или колонии непосредственно в качестве матрицы для ПЦР. Прямой праймер (27F, 5′-AGRGTTTGATCMTGGCTCAG-3′; SEQ ID NO: 1) и обратный праймер (1492R, 5′-GGTTACCTTGTTACGACTT-3′; SEQ ID NO: 2) составляли согласно Singer et al. (2016) с небольшими модификациями. Продукты ПЦР секвенировали непосредственно с помощью прямого и обратного праймеров для ПЦР. Высококачественные отрезки последовательности подвергали BLAST-поиску по базе данных NCBI Nucleotide Collection (nr/nt) для проверки таксономии. Полученные полноразмерные последовательности 16S-рДНК приведены здесь как SEQ ID NOs: 3-24.
Полногеномное секвенирование (WGS). Полногеномное секвенирование биологически чистых изолятов проводили на установке PacBio RSII (Pacific Biosciences, Menlo Park, CA, США) в соответствии с рекомендованным производителем методом получения и секвенирования библиотеки последовательностей. В среднем получали 73 000 прочтений из микробных изолятов длиной в среднем 24 т.н. Сборку генома de novo проводили с помощью иерархического процесса сборки генома (Hierarchical Genome Assembly Process, HGAP; Pacific Biosciences, Menlo Park, CA, США).
Полногеномное выравнивание. Количество известных видов бактерий с полностью секвенированным геномом быстро возросло за последние несколько лет, но все еще остается небольшим по сравнению с количеством последовательностей 16S-рРНК. Однако, когда доступен эталонный геном, то проведение полногеномного выравнивания является одним из самых надежных способов проверки соответствия на уровне вида или даже штамма. Для этого загружали высококачественные микробные геномы из RefSeq и проводили выравнивание с полногеномными последовательностями изолятов с помощью MUMmer (Delcher et al., Nucl. Acids Res. 30: 2478-2483, 2002; Kurtz et al., Genome Biol. 5: R12, 2004), который идентифицирует максимальные уникальные совпадения (“MUMs”) между очень длинными последовательностями.
Идентификация и классификация генов 16S-рРНК и консервативных генов филогенетических маркеров (pMGs). В собранных de novo геномах идентифицировали гены рибосомной РНК (рРНК) с помощью программы Barrnap (Seemann, 2014), которая служит оболочкой для инструмента NHMMer (Wheeler and Eddy, 2013), встроенного в HMMer 3.1. Затем проводили классификацию последовательностей 16S-рРНК с помощью классификатора Naïve Bayesian Classifier из проекта Ribosome Database Project (RDP), а также попарного выравнивания с помощью BLASTn (Camacho et al., BMC Bioinformatics 10:421, 2009; Cole et al., Nucleic Acids Research 42(D1):633-642, 2014).
По мере возрастания числа организмов с секвенированными геномами все более распространенным становится использование этих данных для более точной таксономической классификации. База данных specI содержит последовательности для 40 консервативных генов филогенетических маркеров (pMGs), которые были отобраны из базы данных COG (Conserved Orthologous Groups) (Tatsuov et al., Science 278: 631-637, 1997). Большая часть из них связана с обработкой генетической информации. В базе данных specI имеются последовательности для нескольких тысяч видов бактерий (Mende et al., Nature Methods 10: 881-884, 2013). Поэтому у собранных геномных последовательностей каждого микробного изолята идентифицировали гены, кодирующие белки, с помощью Prodigal (v2.6.2) (Hyatt et al., BMC Bioinformatics 11: 119, 2010), в котором используются эвристические пороги, разработанные в сотрудничестве с экспертами по геномам в Объединенном институте генома Министерства энергетики США (JGI). Транслированные кодирующие последовательности, идентифицированные при помощи Prodigal, аннотировали с помощью программы FetchMG, встроенной в specI (v1.0), в которой для идентификации последовательностей, принадлежащих каждому pMG из COG в пределах данного протеома, используется набор эмпирически определенных отсечек. Затем кодирующие гены, у которых транслированные последовательности были идентифицированы FetchMG как принадлежащие к одному из 40 pMGs в specI, сопоставляли с нетранслированными эталонными pMG из базы данных specI с помощью BLASTn.
Таксономическая классификация микробных изолятов. По традиции определение микроорганизмов на уровне вида зачастую решается на основе одного универсального маркерного гена, гена 16S-рРНК (например, см. Stackebrеt et al., Int. J. Syst. Bacteriol. 44: 846-849, 1994; Janda et al., J. Clin. Microbiol. 45: 2761-2764, 2007). Однако в сообществе микробиологов заметили, что идентичность микроорганизмов, для которых уже доступна полногеномная информация, не всегда коррелирует с идентичностью, определяемой при помощи подходов, обычно применявшихся до прихода высокопроизводительного секвенирования следующего поколения. Более того, полногеномная информация доступна не для всех микроорганизмов, а многие базы данных, в которых зафиксирован генетический ландшафт микроорганизмов, не подвергались проверке/исправлению. Таким образом, отнесение новых изолятов к видам/штаммам микроорганизмов является сложной задачей.
Учитывая вышеописанные ограничения по таксономической идентификации, для отнесения каждого из описанных здесь изолятов к видам микроорганизмов был принят многосторонний подход. Для каждого изолята собирали генетическую информацию типа полногеномного секвенирования, анализа 40 консервативных генов филогенетических маркеров и анализа генов 16S-рРНК. Были заключены контракты с двумя сторонними лабораториями, предлагающими услуги по таксономической идентификации, на проведение идентификации на уровне рода и вида на основе их собственных баз данных и алгоритмов. Кроме того, проводили поиски гомологии по последовательности полноразмерных генов 16S-рРНК для каждого микроорганизма в трех независимых базах данных: базе данных Greengenes (DeSantis et al., Appl. Environ. Microbiol. 72:5069-5072, 2006), базе данных EZTaxon (Kim et al., Int. J. Syst. Evol. Microbiol. 62:716-721, 2012) и базе данных Национального центра информации по биотехнологии (U.S. National Library of Medicine, Bethesda, MD).
Каждый изолят относили к какому-то виду, если по меньшей мере три и более поиска в базах данных (включая собственные базы данных третьих сторон и общедоступные базы данных, приведенные выше) и службы по идентификации давали идентичные определения вида. По меньшей мере для трех изолятов поиск по базам данных не дал идентичного определения вида по меньшей мере в трех базах данных. В этих случаях принимали решение по здравому суждению (Bacillus sp., Pseudomonas sp. и Paenibacillus lautus).
Исходя из вышесказанного, идентификация микроорганизмов проводилась следующим образом.
• Lactobacillus delbrueckii. Результаты всех анализов убедительно подтверждают идентификацию этого изолята как Lactobacillus delbrueckii subsp. bulgaricus ND02.
• Virgibacillus halophilus. Результаты BLASTn по 16S свидетельствуют, что этот изолят может быть штаммом Virgibacillus halophilus, хотя это нельзя проверить другими методами из-за отсутствия подходящих эталонных последовательностей. Результаты по BLASTn 16S соответствуют отнесению на уровне рода по Naïve Bayesian Classifier (NBC), но сами по себе недостаточны для подтверждения классификации на уровне вида.
• Azotobacter vinelandii. Этот изолят может быть новым штаммом Azotobacter vinelandii. Это подтверждается анализом 16S-рРНК и других генов филогенетических маркеров, а также полногеномным выравниванием с двумя ближайшими родственными штаммами. Большое количество как небольших, так и больших отличий, наблюдаемых при полногеномном выравнивании, а также многочисленные небольшие отличия между 16S и кодирующими последовательностями свидетельствуют о том, что это может быть новый штамм, а возможно даже новый вид, хотя этот изолят явно более родствен Azotobacter vinelandii, чем любым другим видам, содержащимся в базах данных.
• Clostridium pasteurianum. Анализ 16S и полногеномное выравнивание подтверждают идентификацию этого изолята как нового штамма Clostridium pasteurianum. База данных specI не содержит никаких эталонных последовательностей для C. pasteurianum, хотя результаты по specI дают дальнейшее подтверждение вплоть до уровня рода.
• Paenibacillus chibensis. Самым убедительным доказательством положительной идентификации этого изолята была классификация на уровне рода согласно RDP NBC, по которой он отнесен к Paenibacillus с вероятностью 1,0. Результаты по 16S и BLASTn по specI подтверждают это отнесение. Точно совпадающего эталонного генома не нашлось в RefSeq, хотя наблюдалась последовательная синтения между собранным геномом и эталонной последовательностью для Paenibacillus sp. Y412MC10. Выравнивание последовательности полноразмерного гена 16S-рРНК в доступных базах данных в настоящее время свидетельствует, что этот изолят принадлежит к Paenibacillus chibensis.
• Streptomyces griseus. Этот изолят явно представляет собой штамм Streptomyces griseus, чрезвычайно близкородственный к NBRC 13350. Это подтверждается анализом генов 16S-рРНК и других филогенетических маркеров, а также полногеномным выравниванием с двумя ближайшими родственными штаммами. Однако полногеномное выравнивание выявило ряд отличий, разбросанных по всему геному, свидетельствуя о том, что этот изолят следует классифицировать как отдельный штамм.
• Pseudomonas sp. Этот изолят явно принадлежит к роду Pseudomonas и очень близкородствен Pseudomonas entomophila, но он не полностью совпадает со штаммом L48, единственным штаммом Pseudomonas entomophila с доступным эталонным геномом. Этот изолят также близкородствен к P. putida и P. fluorescens.
• Pseudomonas putida. Результаты некоторых анализов подтверждают идентификацию этого изолята как Pseudomonas putida. Хотя этот изолят явно чрезвычайно близкородствен штамму P. putida NBRC 14164, наилучшему доступному эталону, однако полногеномный анализ выявил многочисленные небольшие отличия по всему геному, свидетельствуя, что этот изолят следует классифицировать как отдельный штамм.
• Oceanobacillus oncorhynchi. Анализ 16S и полногеномное выравнивание подтверждают идентификацию этого изолята как нового штамма Oceanobacillus oncorhynchi. База данных specI не содержит никаких эталонных последовательностей для O. oncorhynchi, хотя результаты по specI дают дальнейшее подтверждение вплоть до уровня рода.
• Paenibacillus lautus. Результаты всех анализов убедительно подтверждают идентификацию этого изолята как штамма Paenibacillus, близкородственного, хотя и не идентичного хорошо изученному штамму Y412MC10. Учитывая высокую плотность небольших отличий по всему геному при полногеномном выравнивании с Y412MC10, многочисленные небольшие отличия, отмеченные при попарном выравнивании с маркерными генами у Y412MC10, и одинаково хорошее выравнивание с последовательностями штамма HGF5, и тот факт, что Paenibacillus sp. Y412MC10 пока официально не поименован. Выравнивание полноразмерной последовательности гена 16S-рРНК в доступных базах данных в настоящее время свидетельствует о том, что этот изолят принадлежит к виду Paenibacillus lautus.
• Bacillus licheniformis. Результаты всех анализов убедительно подтверждают идентификацию этого изолята как Bacillus licheniformis DSM 13 = ATCC 14580, возможно, вплоть до уровня штамма. Однако этот изолят может оказаться уникальным штаммом, исходя из довольно сильной перестройки и других небольших отличий, выявленных при полногеномном выравнивании, а также точечных мутаций, отмеченных по результатам 16S и specI, хотя такая высокая степень идентичности последовательностей между двумя геномами редко наблюдается, даже у одного и того же вида.
• Lactobacillus vini. Bergey’s Manual of Systematic Bacteriology не содержит никакой дополнительной информации о Lactobacillus mobilis или о потенциально противоположном и столь же плохо изученном Lactobacillus vini (Passoth et al., Microbiology 73: 4354-4356, 2007). Полногеномное выравнивание с единственными доступными эталонами для L. vini указывает на большое число гомологичных участков, но чрезвычайная фрагментация эталонных последовательностей затрудняет оценку взаимосвязи между ними и этим изолятом. Выравнивание полноразмерной последовательности гена 16S-рРНК в доступных базах данных в настоящее время свидетельствует о том, что этот изолят принадлежит к виду Lactobacillus vini.
• Paenibacillus cookii. Самым убедительным доказательством для положительной идентификации этого изолята является классификация на уровне рода согласно RDP NBC, по которой он отнесен к Paenibacillus с вероятностью 1,0. Результаты по 16S и BLASTn по specI подтверждают это отнесение. Точно совпадающего эталонного генома не нашлось в RefSeq, хотя наблюдалась последовательная синтения между собранным геномом изолята и эталонной последовательностью для Paenibacillus sp. Y412MC10. Выравнивание последовательности полноразмерного гена 16S-рРНК в доступных базах данных в настоящее время свидетельствует, что этот изолят принадлежит к виду Paenibacillus cookii.
• Lactobacillus buchneri. Результаты всех анализов убедительно подтверждают идентификацию этого изолята как Lactobacillus buchneri, хотя результаты по specI не подтверждают анализы 16S и полногеномного выравнивания, предположительно потому, что в базе данных отсутствуют необходимые эталонные последовательности. Хотя этот образец явно чрезвычайно близкородствен к штамму L. buchneri CD034, наилучшему доступному эталону, однако полногеномный анализ выявил десятки отличий, включая две большие делеции, свидетельствуя о том, что этот изолят следует классифицировать как отдельный штамм.
• Bacillus megaterium. Результаты всех анализов убедительно подтверждают идентификацию этого изолята как Bacillus megaterium. Хотя этот изолят явно чрезвычайно близкородствен к штамму B. megaterium DSM319, наилучшему доступному эталону, однако полногеномный анализ выявил множество небольших отличий по всему геному, свидетельствуя о том, что этот изолят следует классифицировать как отдельный штамм.
• Acetobacter pasteurianus. Результаты всех анализов убедительно подтверждают идентификацию этого изолята как Acetobacter pasteurianus. Этот изолят явно чрезвычайно близкородствен к штамму A. pasteurianus IFO 3283, наилучшему доступному эталону, но полногеномный анализ выявил много небольших отличий по всему геному даже для самого близкого соответствия, свидетельствуя о том, что этот изолят следует классифицировать как отдельный штамм.
• Clostridium beijerinckii. Результаты всех анализов убедительно подтверждают идентификацию этого изолята как Clostridium beijerinckii. Хотя он явно чрезвычайно близкородствен к штамму C. beijerinckii NCIMB 8052, наилучшему доступному эталону, однако полногеномный анализ выявил много небольших отличий по всему геному, свидетельствуя о том, что этот изолят следует классифицировать как отдельный штамм.
• Lactobacillus casei/paracasei. Результаты всех анализов убедительно подтверждают идентификацию этого изолята как Lactobacillus casei или Lactobacillus paracasei. Наилучшие совпадения по всем анализам последовательно были с представителями этих двух перекрывающихся видов, хотя при всех анализах отмечалось много небольших отличий, охватывающих всю длину генома, свидетельствуя о том, что этот изолят следует классифицировать как отдельный штамм.
• Bacillus flexus. Самым убедительным доказательством для положительной идентификации этого изолята является классификация на уровне рода согласно RDP NBC, по которой он отнесен к Bacillus с вероятностью 1,0. Результаты по 16S и BLASTn по specI, а также полногеномное выравнивание по MUMmer подтверждает это отнесение вплоть до уровня рода. Выравнивание полноразмерной последовательности гена 16S-рРНК в доступных базах данных в настоящее время свидетельствует, что этот изолят принадлежит к виду Bacillus flexus.
• Bacillus sp. Анализы 16S полностью подтверждают идентификацию этого изолята как штамма Bacillus, но ни один из анализов не смог положительно отнести его к поименованному виду. Результаты по 16S и BLASTn свидетельствуют, что это может быть штамм Bacillus kochii, но отсутствие полногеномных последовательностей в доступных базах данных не позволяет проверить это.
• Bacillus subtilis. Результаты всех анализов убедительно подтверждают идентификацию этого изолята как Bacillus subtilis. Таксономия и номенклатура для этого рода и вида довольно сложна, хотя по результатам всех анализов сильно преобладали штаммы, формально классифицированные как B. subtilis. Несмотря на обилие данных по последовательности и многие точные совпадения с отдельными генами, не удалось установить точного совпадения по всему геному, что убедительно указывает на то, что этот изолят является новым штаммом B. subtilis.
• Bacillus amyloliquefaciens. Результаты всех анализов убедительно подтверждают идентификацию как Bacillus amyloliquefaciens subsp. plantarum, хотя таксономия для этого вида в настоящее время довольно сложна. Bacillus amyloliquefaciens subsp. plantarum совсем недавно переименовали в Bacillus velezensis (Dunlap et al., Int. J. System. Evol. Microbiol. 65: 2104-2109, 2015; Dunlap et al., Int. J. System. Evol. Microbiol. 66: 1212-1217, 2015). Эти изменения привели к частичной реклассификации и реорганизации в RefSeq, хотя идентификаторы последовательностей в базах данных specI и RDP по-прежнему указывают на Bacillus amyloliquefaciens subsp. plantarum.
Пример 2. Индивидуальное и совместное культивирование микроорганизмов
Приготовление питательных сред. Исследуемые питательные среды готовили из готовых солей и реагентов. Жидкие среды стерилизовали автоклавированием при 121°C, 15 psi, в течение по меньшей мере 30 минут или фильтрованием через фильтровальные мембраны на 0,45 мкм.
Исследование динамики роста индивидуальных штаммов в монокультурах. Анализировали скорость роста каждого биологически чистого изолята микроорганизмов. Для каждого изолята готовили до 3 мл жидких культур, начиная с матричных чашек. Каждый изолят культивировали 1-3 дня при 30°C, получая культуры с достаточной плотностью в логарифмической фазе. Затем каждый изолят субкультивировали в 24-луночных планшетах, содержащих 1 мл ростовой среды (см. ниже), при оптической плотности (OD600) ~0,05. При помощи Cytation 5 Imaging Reader (BioTek, Winooski, VT, USA) регистрировали профили роста каждого изолята в режиме реального времени (каждые 30 сек) в течение 1 недели в аэробных условиях при 30°C. В качестве отрицательного контроля использовали стерильную среду.
Совместное культивирование микроорганизмов в экспериментах по ферментации
Биореакторы с непрерывным перемешиванием. Ферментацию проводили в биореакторах DASGIP на 2 л (Eppendorf North America, Hauppauge, NY) с рабочим объемом 1,5 л. Сосуды стерилизировали автоклавированием при 121°C, 15 psi в течение 1 часа. Среду для культивирования стерилизировали вместе с сосудами, за исключением PBS, который стерилизировали отдельно и добавляли в биореактор асептически перед инокуляцией. В качестве противопенного средства использовали полипропиленгликоль 2000. Его добавляли в биореактор асептически перед инокуляцией в концентрации 0,07% об/об. Сосуды инокулировали непосредственно из размороженных рабочих флаконов с клетками при объеме инокулята 0,1-0,4% об/об. Температуру при ферментации поддерживали при 30°C. В зависимости от ферментации концентрацию растворенного кислорода иногда контролировали, а иногда нет. Когда её контролировали, то концентрацию растворенного кислорода устанавливали в диапазоне 1-25% от насыщения воздухом. Контроль растворенного кислорода осуществляли с помощью каскада перемешивания, а затем подачи воздуха для поддержания заданного значения. А когда контроль растворенного кислорода не проводился, то встряхивание фиксировали на уровне от 200 до 800 об/мин, а скорость подачи воздуха фиксировали на уровне от 0,1 до 0,5 объемных долей/мин (vvm). Точно так же значение pH ферментационного бульона иногда контролировали, а иногда нет. Когда его контролировали, то значение pH устанавливали в диапазоне от 6,3 до 6,9. Для доведения pH использовали 20% масс. KOH и 13,8% масс. H3PO4. Значение pH ферментационного бульона ежедневно проверяли с помощью автономного pH-метра с тем, чтобы корректировать любые дрейфы у pH-зонда в биореакторе.
Биореактор типа вращающихся колб. Микробный инокулят смешивали с суспензией, содержащей 5,5% масс. белка молочной сыворотки и 1,2% масс. йогурта в воде (“чан C”), и суспензией, содержащей 0,1% масс. спирулины и 0,1% масс. экстракта ламинарии в воде (“чан A”). Суспензии в чан A и чан C готовили индивидуально за 3 дня до смешивания с посевной культурой и инкубировали при обычной температуре. Посевную культуру, чан C и чан A смешивали в пропорции 81:9:9. После перемешивания эту смесь смешивали с суспензией дополнительных компонентов, содержащей 70% об. патоки, 0,5% об. HYT B, 0,003% вес/об. гуммиарабика и 0,02% вес/об. пивных дрожжей (S. cerevisiae), и дополнительной водой в соотношении 16:34:50. Смесь ферментировали в течение 7 дней при обычной температуре (19-35°C). Через 7 дней колбы (1,7 л) аэрировали в течение 30 мин через каждый день. Добавляли еще воды (примерно на 10% об.) и продолжали ферментацию в тех же условиях еще 10 дней. Добавляли еще воды (примерно на 4% об.) и продолжали ферментацию в течение еще 7 дней, после чего отбирали образцы для анализа.
Анализ микробной популяции – капельный цифровой метод ПЦР. Система капельной цифровой ПЦР (ddPCR) обеспечивает обнаружение и количественное определение присутствия целевых организмов (Dreo et al., Anal. Bioanal. Chem. 406: 6513-6528, 2014; Yin et. al., Journal of Microbiological Methods 65: 21-31, 2006). Праймеры, специфичные для 22 видов бактерий, идентифицированных в Примере 1, составляли, используя уникальные последовательности из генов 16S и/или уникальные последовательности кодирующих генов, идентифицированных при сборке генома для WGS (таблица 2 и 3). ddPCR проводили с использованием EvaGreen Supermix либо Supermix for Probes (BioRad, Hercules, CA, USA) в соответствии с рекомендациями производителя на установке QX200 Droplet Digital PCR System (BioRad, Hercules, CA, USA), а затем данные ddPCR анализировали в QuantaSoft (BioRad, Hercules, CA, USA).
Таблица 2. Прямые и обратные праймеры для метода ddPCR с EvaGreen Supermix
pasteurianus
(SEQ ID NO: 25)
vinelandii
(SEQ ID NO: 27)
(SEQ ID NO: 29)
flexus
licheniformis
(SEQ ID NO: 33)
megaterium
(SEQ ID NO: 35)
AAAGATGG (SEQ ID NO: 37)
subtilis
(SEQ ID NO: 39)
beijerinckii
(SEQ ID NO: 41)
pasteurianum
(SEQ ID NO: 43)
buchneri
(SEQ ID NO: 45)
delbrueckii
(SEQ ID NO: 47)
paracasei (casei)
(SEQ ID NO: 49)
vini
(SEQ ID NO: 51)
oncorhynchi
(SEQ ID NO: 53)
lautus
chibensis
TG (SEQ ID NO: 57)
cookii
(SEQ ID NO: 59)
(SEQ ID NO: 61)
putida
(SEQ ID NO: 65)
(SEQ ID NO: 67)
CCTGTT (SEQ ID NO: 68)
halophilus
(SEQ ID NO: 69)
Таблица 3. Олигонуклеотиды для метода ddPCR с Supermix for Probes
Разработка среды для монокультур. Каждый из микробов выделяли на различных средах. Проверяли, будет ли достаточно одного состава среды для роста всех 22 изолятов.
Сначала проверяли рост всех 22 изолятов на одной патоке. Оказалось, что 5 из 22 штаммов проявляли заметный рост. Затем состав среды оптимизировали со включением основных элементов типа фосфатов, натрия, калия и хлорида (в виде коммерчески доступного фосфатно-солевого буфера (PBS). Это приводило к росту еще 6 изолятов. Затем добавляли источники аминокислот, азота и пептидов/белков в виде порошка молочной сыворотки пищевого уровня и полученного энзиматически соевого экстракта без ГМО (Ferti-Nitro Plus Plant N; Ferti-Organic, Brownsville, TX, USA). Одна только сыворотка не улучшала существенно показатели роста большинства изолятов, за исключением Bacillus sp. Ferti-Nitro Plus Plant N сам по себе явно стимулирует рост большого числа микробов. А вместе с белком молочной сыворотки Ferti-Nitro Plus Plant N давал умеренный синергический эффект, еще больше стимулируя рост A. pasteurianus, O. oncorhynchi, C. pasteurianum, L. delbrueckii и V. halophilus. Результаты представлены в таблице 4.
Таблица 4. Оценка показателей роста отдельных микроорганизмов в среде PBS с патокой
в воде (% вес/об.)
(% вес/об.)
(-) Рост не выявляется; (+) слабый рост; (++) умеренный рост; (+++) сильный рост
(*) Изоляты культивировали в анаэробных условиях
В других экспериментах определяли среду, которая бы поддерживала рост двух изолятов – C. pasteurianum и Azotobacter vinelandii. Кроме того, стремились разработать среду, которая бы улучшала рост некоторых изолятов типа O.oncorhynchi и V. halophilus, L. delbrueckii и L. vini, которые не проявляли устойчивого роста в среде, приведенной выше.
Используя информацию об оптимальных условиях роста для каждого изолята (Bergey’s Manual of Systematics of Archaea and Bacteria; Ed., William B. Whitman), вышеприведенные данные, общедоступные сведения об элементном составе бактерий (Ritt-mann and McCarty. Environmental Biotechnology: Principles and Applications; 2001 ISBN 0072345535), и рекомендованную среду для культивирования Azotobacter (1713 Modified Azotobacter Medium I; ATCC), тестировали различные составы сред на рост 22 изолятов микроорганизмов в монокультурах. Результаты представлены в таблице 5.
В этих экспериментах тестировали фосфат калия в качестве источника элементарного фосфора и калия вместе с различными концентрациями хлорида натрия (чтобы удовлетворить потребности таких галофильных микроорганизмов, как O. oncorhynchi и V. halophilus), а также молочную сыворотку и Ferti-Nitro Plus Plant N в качестве источника (без ограничения) аминокислот и азота. Наряду с патокой, в специально разработанную смесь солей добавляли микроэлементы, как описано ниже.
Таблица 5. Оценка показателей роста отдельных микроорганизмов в среде AAM01 с патокой
(% вес/об.)
(**) AAM01 (среда Agrinos для Azotobacter 01) состоит из сульфата железа (0,12 г/л); сульфата магния (0,3 г/л); хлорида кальция (0,1 г/л); хлорида марганца (0,001 г/л), молибдата натрия (0,001 г/л); сульфата калия лимонной кислоты (0,12 г/л)
(*) Изоляты культивировали в анаэробных условиях
(-) Рост не выявляется; (+) слабый рост; (++) умеренный рост; (+++) сильный рост
В этих экспериментах Azotobacter vinelandii и C. pasteurianum показали хорошие результаты. Многие другие изоляты также хорошо росли, однако, в отличие от предыдущего состава (например, PBS с патокой), не все штаммы росли устойчиво в одной среде.
В других экспериментах оценивали рост отдельных изолятов микроорганизмов в среде, которая включала дрожжевой порошок (0,021 г/л; YP), гуммиарабик (0,028 г/л; AG), черную патоку, порошок молочной сыворотки (1,56 г/л); WP), спирулину (0,029 г/л, SP), экстракт ламинарии (0,029 г/л; KE) и 0,8% вес/об. NaCl (так как есть данные о том, что такие изоляты, как O. oncorhynchi и V. halophilus, лучше растут в присутствии этой соли). Эта среда именуется здесь как среда “Экстракт”. Состав микроорганизмов отслеживали по времени с помощью ddPCR, как описано выше. Выявленные микроорганизмы отмечали как (+). Микроорганизмы, которые были ниже предела обнаружения (BDL) метода ddPCR, отмечали как (-). Результаты этого эксперимента представлены в таблице 6.
Таблица 6. Оценка показателей роста отдельных микроорганизмов в среде “Экстракт”
* Культивировали в анаэробных условиях
Исследовали эффективность ферментации в биореакторах небольшого объема с двумя составами среды. Первая среда состояла из патоки, PBS, белка молочной сыворотки и Ferti-Nitro Plus Plant N (таблица 4). В отличие от сред на основе химических веществ, используемых при ферментации Azotobacter в лабораторных объемах (типа AAM01), не было солей с потенциальными проблемами безопасности. Многие составы оптимальных сред для роста микроорганизмов исследовательского уровня рекомендуют использовать такие микроэлементы, как селен и молибден (который является мощным кофактором ферментов, особенно для нитрогеназ). Среда “Экстракт” (таблица 6), состоящая из патоки и других натуральных экстрактов, очень похожа на среды, которые сейчас применяются.
Совместное культивирование в экспериментах по ферментации в небольших объемах. Для изучения условий ферментации, которые бы поддерживали совместное культивирование всех 22 штаммов микроорганизмов, использовали настольные ферментеры. Полученные продукты ферментации анализировали на наличие и численность каждого микроба через 4-28 дней после инокуляции. Использовали два типа конфигурации биореакторов для ферментации.
Биореакторы с непрерывным перемешиванием. Первая конфигурация биореактора – реактор с непрерывным перемешиванием ёмкости (CSTR). В биореакторы DASGIP на 2 л, содержащие 1,5 л среды, инокулировали 22 ODs микроорганизмов (по 1 OD для каждого отдельного штамма). В этих экспериментах использовали среду на основе результатов из табл. 3, которая содержала следующие ингредиенты: фосфатно-солевой буфер (1х), черную патоку (2-10% вес/об.), белки молочной сыворотки (0,1-0,5% вес/об.), Ferti-Nitro Plus Plant N (0,25-1,25% вес/об.) с экстрактом ламинарии (0,0067%) или без него, дрожжевой порошок (0,0033% вес/об.) и/или спирулины (0,0067% вес/об.). В этих экспериментах варьировали состав газов (от 5 до 21% O2; от 95 до 79% N2), скорость потока газа (от 5 до 45 стандартных л/час), pH (от 6,3 до 6,7), перемешивание (200-1100 об/мин) и растворенный кислород (без контроля до 25%). Температура оставалась постоянной при 30°C. Количественный анализ и анализ состава микроорганизмов под конец ферментации определяли методом ddPCR с помощью штамм-специфичных зондов, как описано выше. Выявленные микроорганизмы отмечали как (+). Микроорганизмы, которые были ниже предела обнаружения (BDL) метода ddPCR, так и отмечали (BDL). Типичные результаты представлены в таблице 7.
Таблица 7. Сводный профиль микроорганизмов при совместной ферментации с использованием CSTR на 1,5 л
патоки
патоки
(*) BDL = ниже предела обнаружения
В других экспериментах микроорганизмы, которые не выявлялись при совместном культивировании всех 22 штаммов (см. выше), тестировали, чтобы определить, могут ли они расти вместе. Для этого проверяли влияние последовательности инокуляции данных микроорганизмов (все вместе либо сначала анаэробы либо сначала аэробы), а также уровня растворенного кислорода во время инокуляции и при ферментации, а также состава среды. Уровень растворенного кислорода контролировали путем изменения подачи газа по скорости потока и/или перемешивания. В таблице 8 представлены результаты по совместному культивированию C. beijerinckii (строгий анаэроб), S. griseus и B. flexus (строгие аэробы). Как и в предыдущих экспериментах, детектирование и оценку роста микроорганизмов проводили методом ddPCR.
Таблица 8. Сводка по схеме эксперимента и профилю микроорганизмов при совместном культивировании C. beijerinckii, S. griseus и B. flexus
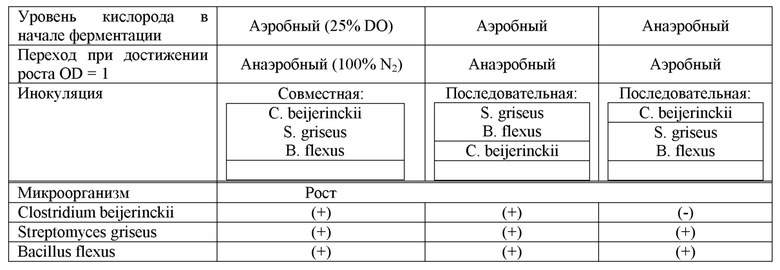
Биореактор типа вращающихся колб. Инокулят, содержащий все 22 микроба, разбавляли средой “Экстракт” и проводили ферментацию патоки во вращающихся колбах, как описано выше. Состав микроорганизмов отслеживали по времени методом ddPCR. Выявленные микроорганизмы отмечали как (+). Микроорганизмы, которые были ниже предела обнаружения (BDL) метода ddPCR, так и отмечали (BDL). Результаты эксперимента представлены в таблице 9.
Таблица 9. Сводка по микробному профилю при совместном культивировании 22 микроорганизмов во вращающихся колбах на протяжении 28 дней
BDL = ниже предела обнаружения
Совместное культивирование в ферментерах промышленного масштаба. Исходя из потребностей бактерий при оптимальном совместном культивировании и требуемого конечного качества микробиологической консорции (оцениваемого по ddPCR), в ферментеры объемом до 300 л инокулировали аэробные и/или анаэробные бактерии из описанных выше групп. Конечное значение OD600 при инокуляции каждого штамма рассчитывали так, чтобы оно составляло от 6,67×10−5 до 6,67×10−4. Среда для ферментации включала патоку, белки молочной сыворотки, экстракт ламинарии, дрожжевой порошок, спирулину, гуммиарабик и хлорид натрия. Для поддержания рН между 5,2 и 7,2 в качестве щелочного и кислого раствора использовали гидроксид аммония и фосфорную кислоту, соответственно. Температуру контролировали между 28°C и 35°C, а растворенный кислород поддерживали между 0% и 25% на всем протяжении ферментации (вплоть до 3 дней). Усредненный состав микроорганизмов в продуктах ферментации представлен в таблице 10.
Таблица 10. Усредненный состав микроорганизмов в продуктах ферментации
n = 16, анаэробные; n = 10, аэробные
BDL = ниже предела обнаружения
Анализ жизнеспособной микробной нагрузки методом нанесения на чашки в аэробных и анаэробных условиях. Анализ количества микроорганизмов проводили методом нанесения на чашки для определения колониеобразующих единиц (к.о.е.) в образцах. Все образцы хранили при комнатной температуре в светонепроницаемых и герметичных контейнерах. После энергичного перемешивания образца для равномерного распределения содержимого сохраняли 1 мл. Из этой аликвоты в асептических условиях отбирали 0,1 мл и смешивали с 9,9 мл стерильной воды в культуральной пробирке (разведение 10−2). Затем пробирки перемешивали на вибромешалке (например, 60 секунд при 2000 об/мин) и готовили 10-кратные серийные разведения в воде (вплоть до 1:109). После этого по 100 мкл каждого разведения размазывали по полутвердой среде в чашках Петри на 100 мм с помощью стерильного L-образного шпателя. Использовали чашки, содержащие стандартные промышленные среды типа агара для стандартных методов (SMA; BD № 247940) или питательного агара (NA; BD № 213000). Затем инокулированные чашки инкубировали в камерах с контролируемой температурой от 22°C до 35°C. Для определения числа анаэробных микробов чашки сначала помещали в анаэробные боксы (например, BD GasPak™ EZ Container Systems, BD Diagnostics), а затем инкубировали при нужной температуре. В некоторых случаях исследуемые аликвоты сначала инкубировали в стерильной воде с пептоном на протяжении до 3 дней при температурах до 35°C, а затем готовили серийные разведения и посев на чашки, как описано выше. После инкубации подсчитывали все колонии на отобранных чашках с помощью счетчика колоний типа темнопольного счетчика колоний Quebec® (Reichert) и рассчитывали к.о.е./мл. При посеве на чашках появлялось до 1,1E+09 к.о.е./мл в аэробных и анаэробных условиях.
Пример 3. Идентификация потенциальной метаболической активности микроорганизмов
Анализ солеустойчивости. Все штаммы в микробиологической консорции проявляли приемлемый рост в лабораторной среде типа дрожжевой экстракт-пептон-декстроза (YPD, Difco 242820). Поэтому эту среду использовали для определения солеустойчивости каждого изолята при различных количествах хлорида натрия (стандарта для испытания на солеустойчивость) вплоть до 5% вес/об. Каждый изолят культивировали в 2 мл среды в идеальных условиях роста: 30°C с перемешиванием (125-175 об/мин) для аэробов и 35°C без перемешивания для анаэробов в анаэробных камерах (BD Diagnostics). Через 24, 48 и 72 часа регистрировали рост для каждой культуры на основе общей эквивалентности со стандартами MacFarland (доступны во всемирной паутине по адресу pro-lab.com/inserts/McFarland.pdf). Любые изоляты, проявляющие рост при 5% NaCl через 72 часа, отмечали как солеустойчивые к NaCl ≥5%, рост при 2,5% – как ≥2,5% и так далее.
Анализ фиксации азота (N). Готовили и стерилизовали в автоклаве безазотную полутвердую среду, содержащую сахарозу 5,0 г/л, MgSO4 0,2 г/л; KH2PO4 0,8 г/л; FeSO4 0,04 г/л; Na2MoO4 0,005 г/л; CaCO3 2 г/л и 15 г/л агара. Из матричной культуральной чашки переносили одну колонию каждого изолята в лишенную N чашку в асептических условиях. Колонии рассеивали по свежим лишенным N чашкам методом штрихования. Условия роста варьировали в зависимости от изолята: анаэробные/микроаэрофильные бактерии инкубировали при 35°C в анаэробных камерах, а аэробные бактерии инкубировали при 30°C. Время инкубации составляло до 1 недели, пока не отмечался рост. Положительными считались только сильные азотфикаторы (например, обильный рост).
Анализ солюбилизации соли кальция. Готовили и стерилизовали в автоклаве полутвердую среду с карбонатом кальция, содержащую MgSO4 0,3 г/л; CaCl2 0,1 г/л; FeSO4 0,12 г/л; K2SO4 1 г/л; сахарозу 20 г/л; Na2MoO4 0,01 г/л; MnCl2 0,01 г/л; дрожжевой экстракт 5 г/л; пептон 10 г/л; CaCO3 2 г/л; агар 15 г/л. Из матричной культуральной чашки переносили одну колонию каждого изолята в карбонатную чашку в асептических условиях. Проводили единственный штрих из середины чашки. Условия роста варьировали в зависимости от изолята: анаэробные/микроаэрофильные бактерии инкубировали при 35°C в анаэробных камерах, а аэробные бактерии инкубировали при 30°C. Время инкубации составляло до 1 недели, пока не отмечалось наличие зоны просветления, прилегающей к бактериям. Положительным считалось только явное просветление осадка CaCO3.
Анализ солюбилизации фосфатной соли. Готовили и стерилизовали в автоклаве полутвердую среду с фосфатом кальция, содержащую MgSO4 0,3 г/л; CaCl2 0,1 г/л; FeSO4 0,12 г/л; K2SO4 1 г/л; сахарозу 20 г/л; Na2MoO4 0,01 г/л; MnCl2 0,01 г/л; дрожжевой экстракт 5 г/л; пептон 10 г/л; Ca3(PO4)2 5 г/л; агар 15 г/л. Из матричной культуральной чашки переносили одну колонию каждого изолята в фосфатную чашку в асептических условиях. Проводили единственный штрих из середины чашки. Условия роста варьировали в зависимости от изолята: анаэробные/микроаэрофильные бактерии инкубировали при 35°C в анаэробных камерах, а аэробные бактерии инкубировали при 30°C. Время инкубации составляло вплоть до 1 недели, пока не отмечалось наличие зоны просветления, прилегающей к бактериям. Положительным считалось только явное просветление осадка фосфата кальция.
Анализ солюбилизации солей цинка. Для проверки штаммов на способность к солюбилизации цинка использовали при анализе четыре типа цинка: Zn5(CO3)2(OH)6 (карбонат-гидроксид цинка), Zn3(PO4)2 (фосфат цинка), ZnO (оксид цинка) и ZnSO4 (сульфат цинка). Каждый тип цинка добавляли при 0,2% (вес/об.) в агаровую среду типа YPD или бульона из мозга и сердца (BHI). Для каждого штамма затем отбирали одну колонию и наносили штрихом на обе полутвердые среды в чашках Петри. Аэробы инкубировали при 30°C, а анаэробы инкубировали при 35°C в статическом инкубаторе в течение 3 дней. Через 24, 48 и 72 часа чашки проверяли на наличие признаков просветления в среде, что интерпретируется как положительный показатель солюбилизации цинка.
Анализ мобилизации железа. Целый ряд почвенных микроорганизмов продуцирует так называемые сидерофоры в средах с низкой концентрацией железа – необходимого микронутриента. Эти соединения образуют водорастворимые комплексы с Fe3+, которые могут высвобождаться при дефиците железа. Сидерофоры бактерий приносят пользу как растениям, так и микроорганизмам в качестве локализованного источника железа. Проводили анализ полногеномных последовательностей для каждого из 22 изолятов с целью выявления генов, кодирующих сидерофоры, пути биосинтеза сидерофоров, а также такие рецепторы и переносчики сидерофоров, как Yus, Yfh, Yfi, Asb, Fur, TonB, ExbB, ExbD, цитрат, десферриоксамин, десфероксамин, феррихром, фузаринин, орнибактин, хризобактин, вибриобактин, микобактин, пиовердин, пиохелин, йерсиниабактин, энтеробактин, ахромобактин, акинетобактин, азотобактин, бациллибактин и ангвибактин. Это позволило определить, обладает ли данный микроорганизм метаболическим арсеналом для продукции и/или транспорта соединений, мобилизующих железо.
Анализ способности к дефосфорилированию органических веществ. Бактерии могут выделять целый ряд микробных ферментов, которые при воздействии на органические вещества могут образовывать формы фосфата, доступные для растений. Эти ферменты включают неспецифические фосфатазы, которые дефосфорилируют фосфоэфирные и/или фосфоангидридные связи в органическом веществе, фитазы, которые высвобождают фосфор из фитиновой кислоты, фосфонатазы и C–P-лиазы, которые диссоциируют связи C–P в фосфорорганических соединениях. Проводили анализ полногеномных последовательностей для каждого из 22 изолятов с целью выявления генов, кодирующих эти ферменты в функциональных метаболических путях. Это позволило определить, обладает ли данный микроорганизм арсеналом ферментов для дефосфорилирования органических веществ в почве.
Определение хитиназы. Определение проводили на чашках с коллоидным хитином в основном, как описано в Hsu and Lockwood (Applied Microbiology, 29: 422-426, 1975). Колонии бактерий (1–3-дневные), отобранные из матричных чашек, рассеивали штрихом на чашки с коллоидным хитином (полусухой хитин, 5 г/л; K2HPO4, 0,7 г/л; KH2PO4, 0,3 г/л; MgSO4·5H2O, 0,5 г/л; FeSO4·7H2O, 0,01 г/л; ZnSO4, 0,001 г/л; MnCl2, 0,001 г/л и агар 15 г/л). Чашки инкубировали до 1 недели при 30°C для аэробов или при 35°C в анаэробных условиях для анаэробов. Положительные по хитиназе изоляты идентифицировали по просветлению в среде и/или по значительному росту микроорганизмов.
Определение целлюлазы. Определение на чашках с дезоксиметилцеллюлозой было адаптировано из Alves et al. (The Open Microbiology Journal, 8:25-31, 2014). Вкратце, колонии бактерий (1–3-дневные), отобранные из матричных чашек, рассеивали штрихом на полутвердую среду (дезоксиметилцеллюлоза, 0,5% вес/об., агароза, 1,5% вес/об., трис-HCl, 50 мМ рН 6,8; CaCl2, 1 мМ). Затем изоляты бактерий инкубировали при 30°C для аэробов и при 35°C в анаэробных условиях для анаэробов вплоть до 1 недели. После этогоё каждую чашку обрабатывали раствором Congo Red (0,25% вес/об. в 0,1 М трис-HCl, рН 8,0) и удаляли краситель с помощью 0,5% NaCl в 0,1 М трис-HCl, рН 8,0. Положительные по целлюлазе изоляты идентифицировали по просветлению в среде и/или по значительному росту микроорганизмов.
Анализ продукции индол-3-уксусной кислоты (IAA). Анализ IAA проводили на каждом микробном изоляте, выращенном в жидкой среде с добавлением 1-5 мг/мл триптофана. Через 7-14 дней после инокуляции культуры тестировали на продукцию IAA с помощью реагента Salkwoski, как описано в Glickmann et al. (Applied and Environmental Microbiology, Feb. 1995, p. 793-796). Вкратце, осветленную отработанную среду из каждой культуры смешивали с реагентом Salkwoski в соотношении 1:1. После 30 мин инкубации измеряли поглощение при 540 нм. Определение продукции IAA проводили по предварительно составленной стандартной кривой с использованием очищенной IAA (Alfa-Aesar, Tewksbury, MA, USA). Кроме того, проводили анализ полногеномных последовательностей для каждого из 22 изолятов с целью выявления путей биосинтеза ауксина.
Другие метаболические анализы. Для дополнительных метаболических активностей, включая анализы денитрификации, продукции уреазы и ассимиляции яблочной кислоты, использовали препараты для идентификации API® фирмы bioMérieux в соответствии с рекомендациями производителя (bioMérieux, Inc., Durham, NC, USA).
Результаты по профилированию ключевых метаболических активностей представлены в таблице 11.
Таблица 11. Метаболические активности изолятов микроорганизмов
организм
subtilis
Пример 4. Оценка активности стимулирования роста растений
Семена огурцов, приобретенные в The Seed Kingdom, проращивали в течение 4 дней при 22-24°C в ростовой камере с контролируемой температурой и влажностью (Sheldon Manufacturing, Inc., Cornelius, OR). Проращивание проводили в рулонах бумаги для проращивания (Anchor Paper, Saint Paul, MN), пропитанной разбавленной смесью жидких удобрений (50 ppm 20-20-20 NPK; Grow More, Gardena, CA) в воде и препаратом Agrinos microbial consortium (AMC) в разведении от 1:1000 до 1:2000. Препарат AMC представляет собой жидкий продукт, полученный путем совместного культивирования 22 микроорганизмов, приведенных в таблице 10, при совместном культивировании в промышленном масштабе, описанном в примере 2.
Затем развернутые и синхронизированные проростки пересаживали в готовую питательную среду типа горшечного грунта (Sunshine Mix), обработанную удобрениями и микробами. Обработка горшечного грунта состояла из разбавленной смеси жидких удобрений (50 ppm NPK) и препарата AMC в разведении от 1:1000 до 1:2000. Для каждой обработки использовали 17-18 растений. Горшки рандомизировали по корзинам в определенных условиях роста: 16-24°C и 12-часовой световой период. Корзины поливали 2-3 раза в неделю с 50 ppm NPK. Через 33 дня измеряли индекс листовой поверхности (LAI) у концов настоящих листьев каждого растения (фиг. 2). Данные анализировали однофакторным методом ANOVA (дисперсионный анализ) для сравнения образцов в эксперименте.
Пример 5. Оценка действия на культуры
В этом примере описаны определенные методы оценки действия представленных композиций на такие культуры, как кукуруза, томаты и капуста. Однако специалистам в данной области должно быть ясно, что для оценки активности композиций также можно использовать способы, которые отличаются от данных конкретных способов.
Анализ на кукурузе (ростовая комната). Семена кукурузы (Alberta Lea Seeds, Albert Lea, MN) вымачивали в воде 4 часа при комнатной температуре перед посадкой в питательную среду типа горшечного грунта или безгрунтовой среды (Sunshine Mix), обработанную удобрением и препаратом AMC следующим образом: модифицированный раствор Hoagland (P, 30,97 м.д.; K, 39,1 м.д.; Ca, 40,0 м.д.; Mg, 14,59 м.д.; S, 20,143 м.д.; Fe, 1,010 м.д.; Cu, 0,019 м.д.; Co, 0,012 м.д.; В, 2,44 м.д.; Mn, 0,494 м.д.; Мо, 0,001 м.д.; и Zn, 0,056 м.д.) и препарат АМС в разведении 1:1000. При посадке семян в грунт добавляли 100 мг препарата мочевины с медленным высвобождением. Лотки с 8-9 горшками инкубировали в темноте 3 дня при 25°C в ростовой камере с контролируемой температурой и влажностью (Sheldon Manufacturing, Inc., Cornelius, OR). Затем растения выращивали в течение 13-16 дней в условиях ростовой камеры: 16-24°C и 12-часовой фотопериод. 2-3 раза в неделю проводили полив модифицированным раствором Hoagland. Впоследствии определяли вес сухих побегов после 4-дневной сушки при 75°C (фиг. 3). Данные анализировали однофакторным методом ANOVA (дисперсионный анализ).
Полевые испытания на томатах. Томаты сорта Sun 6366 высаживали на грядки размером 6×1,8 м с дополнительным поливом при посадке. Перед посадкой поле удобряли по 201 кг N/га, 224 кг/га P2O5 и K2O. К томатам на каждой грядке два раза вносили AMC. Первый раз AMC вносили после посадки на поле. AMC вносили посредством капельного орошения (TTape, расстояние между капельницами 20 см) из расчета 1 л/акр. Второй раз AMC вносили непосредственно в почву через 36 дней после посадки вместе с азотным удобрением в виде UAN 28 из расчета 38 л/акр. Контрольные грядки обрабатывали точно так же, как экспериментальные, за исключением того, что в процессе культивирования не использовали АМС.
В насаждениях томатов было одинаковое количество растений. Помидоры собирали вручную через 2 месяца после последнего внесения АМС. Всего с площади 3,35 м2 в центре грядки отбирали 6 растений. Отмечали урожай и качество томатов (зеленые или красные). Регистрировали их вес и анализировали с помощью XCELSTAT2016.4 (Addinsoft Inc.) (фиг. 4).
Полевые испытания на кукурузе. Кукурузу сорта Mycogen 2H723 (Mycogen Seeds, Indianapolis, IN) высаживали в 4-рядной конфигурации по 75 см между рядами длиной 6,1 м. Перед посадкой поле удобряли по 201 кг N/га, 224 кг/га P2O5 и K2O. На каждый участок два раза вносили AMC. Первый раз AMC вносили после посадки на поле. AMC вносили с помощью ранцевого распылителя из расчета 1 л/акр, а почву поливали водой посредством капельного орошения (TTape, капельницы на 20 см). В этом исследовании вода для орошения была ограничена 75% от нормального уровня. Ограничения на воду вводили на 5 недель для полного расцветания. Второй раз AMC вносили непосредственно в почву через 36 дней после посадки. Во время внесения контрольные грядки кукурузы получали дополнительно азотное удобрение в виде UAN 28 из расчета 38 л/акр = 33 кг N/га, в общей сложности 234 кг N/га на протяжении всего исследования. Участки с АМС не получали дополнительно азота и в итоге получили 201 кг N/га. Почва в районе поля имеет высокое значение pH, в среднем 8,1.
Насаждения кукурузы прореживали до 34 000 растений, чтобы было одинаковое количество растений. Кукурузу убирали кукурузным комбайном через 5 месяцев после посадки. Собирали только 2 центральных ряда с 4-рядных участков, что составляет площадь в 9,3 м2 в центре участка, оставляя много пограничных растений вокруг убранного участка. Зерно кукурузы сушили до 15% влажности в течение нескольких дней и взвешивали. Регистрировали вес зерна и анализировали с помощью XCELSTAT2016.4 (Addinsoft Inc.) (фиг. 5).
Полевые испытания на капусте. Капусту сорта Golden Acres сажали в виде семян на четырехрядных грядках длиной 12 футов и шириной 11 футов. Перед посадкой поле удобряли по 20 галлонов на акр 25-7-0-3 S в два ряда через 16 дюймов от центра грядки. Дополнительно вносили удобрение в качестве междурядной подкормки в виде UAN (32% N) по 10 галлонов на акр, получая 90 фунтов N, 15 фунтов P, 0 фунтов K и 6,5 фунтов S на 1 акр. Препарат AMC вносили из расчета 1 л/акр в борозды при посадке, а в случае 2-й обработки AMC вносили второй раз (1 л/акр) через 42 дня после посадки. Поле поливали по бороздам 4 раза с диапазоном полива от 6,3 до 8,9 акродюймов воды на 1 полив, в общей сложности 29,7 акродюймов. Осадки обеспечивали еще 10,7 дюйма в течение жизни культуры. Поле опрыскивали различными инсектицидами, гербицидами и фунгицидами для защиты капусты от биотических стрессов. Почва имела pH 8,0.
Насаждения капусты имели равное количество растений, а её собирали один раз через 95 дней после посева. Собирали все растения в 2-х средних рядах грядки при эффективной площади участка в 220 кв. футов. Затем кочаны сортировали по категориям размера 24 и 18 и взвешивали. Анализировали сортированный вес и общий вес с помощью XCELSTAT2016.4 (Addinsoft Inc.) (фиг. 6).
Пример 6. Анализ корней
Семена товарных культур типа кукурузы и томатов, без ограничения, проращивали в течение 3 дней при 22-24°C в ростовой камере с контролируемой температурой и влажностью (Sheldon Manufacturing, Inc., Cornelius, OR). Проращивание проводили в рулонах бумаги для проращивания (Anchor Paper, Saint Paul, MN), пропитанной разбавленной смесью жидких удобрений (25-100 ppm 20-20-20 NPK; Grow More, Gardena, CA) в воде и препаратом AMC в разведении от 1:1000 до 1:2000. Развернутые и синхронизированные проростки пересаживали в предварительно обработанную безгрунтовую среду типа песка, каменной ваты, вермикулита или другого инертного матрикса. Предварительная обработка среды роста состоит из разбавленной смеси гидропонного питательного раствора типа полного питательного раствора Hoagland (Hoaglund and Arnon, The Water-Culture Method for Growing Plants Without Soil, 1938) и препарата AMC в разведении от 1:1000 до 1:5000. При каждой обработке по меньшей мере 12 растений, включая контрольные растения, рандомизировали в опорные поддоны. Растения выращивали в стандартных тепличных условиях вплоть до 45 дней. По завершении экспериментов анализировали анатомию и морфологию корней.
Пример 7. Жизнеспособность лиофилизованных микроорганизмов
Лиофилизация. Культивируемые индивидуально или совместно микроорганизмы в оптимальных средах (таблица 12) подвергали сублимационной сушке для оценки жизнеспособности. Вкратце, в монокультурах микроорганизмы культивировали при 30-35°C с постоянным перемешиванием (200 об/мин) в термостатированном инкубаторе в объеме 5 мл культуры в течение 24 часов в анаэробных или аэробных условиях. При совместном культивировании микроорганизмы выращивали в биореакторах DASGIP на 2 л (Eppendorf North America, Hauppauge, NY) со средой, содержащей 2% вес/об. патоки, 0,1% вес/об. порошка молочной сыворотки, 0,25% вес/об. Ferti-Nitro Plus Plant N (Ferti-Organic, Brownsville, TX, USA), 0-4% вес/об. NaCl, 0,02% KCl, 0,115% вес/об. Na2HPO4 и 0,02% KH2PO4, рН 5,7-7,0, при 30-35°C. Анаэробные группы культивировали в отсутствие кислорода.
Для каждой культуры определяли оптическую плотность при 600 нм. Каждую культуру впоследствии смешивали с раствором маннита/лиопротектора (OPS Diagnostics, Lebanon, NJ, USA) в соответствии с рекомендациями производителя, и суспензию микроорганизмов дозировали во флаконы для лиофилизации (OPS Diagnostics, Lebanon, NJ, USA), которые заполняли на 1/3-1/5 объема, а затем снабжали разъемной пробкой. Через 60 минут при -80°C флаконы помещали в систему сублимационной сушки FreeZone 6 (Labconco, 
 Kansas City, MO), применяли вакуум, и вода в образцах сублимировалась в течение ночи (0,04 мБа, -50°C). После сублимационной сушки разъемные пробки опускали во флаконы, дополнительно закрепляли алюминиевой полоской и обжимали. Образцы хранили при 4°C, пока не понадобятся.
Kansas City, MO), применяли вакуум, и вода в образцах сублимировалась в течение ночи (0,04 мБа, -50°C). После сублимационной сушки разъемные пробки опускали во флаконы, дополнительно закрепляли алюминиевой полоской и обжимали. Образцы хранили при 4°C, пока не понадобятся.
Таблица 12. Среды, использовавшиеся для роста отдельных микроорганизмов
YPD: дрожжевой экстракт-пептон-декстроза (BD, # 242720); YPDS: YPD + 8 г/л NaCl; RCM: усиленная среда для клостридий (BD, # 218081); BHI: бульон из мозга и сердца (HiMedia, # LQ077); BHIS: BHI + 45 г/л NaCl; RhX: среда ATCC 111 = среда X для Rhizobium; MRS: MRS для лактобацилл (BD, # 288210); M-HYTA: патока, белки молочной сыворотки, экстракт ламинарии, дрожжевой порошок, спирулина и NaCl.
Для оценки жизнеспосбности каждую лиофилизованную культуру подвергали регидратации и определяли способность к росту. Регидратированные культуры инокулировали в свежие среды при OD600 = 0,1. Отслеживали рост на протяжении 3 дней. В качестве контроля использовали нелиофилизованные культуры. Результаты представлены в таблице 13.
Таблица 13. Рост микроорганизмов после лиофилизации и регидратации
a (+) означает рост после лиофилизации
* Культивировали в анаэробных условиях
Ввиду многих возможных воплощений, к которым могут быть применены принципы изобретения, следует иметь в виду, что представленные воплощения приводятся только для примера и не должны рассматриваться как ограничивающие объем изобретения. А объем изобретения определяется следующей формулой изобретения. Поэтому мы заявляем в качестве изобретения все, что входит в рамки и суть этой формулы.
--->
SEQUENCE LISTING
<110> Agrinos AS
Kendirgi , Frederic
Wagner, D, Ry
Liu, Xing Liang
Yoon, Sung-Yong H.
<120> DEFINED MICROBIAL COMPOSITIONS
<130> 9223-96265-02
<150> US 62/381,441
<151> 2016-08-30
<160> 136
<170> PatentIn version 3.5
<210> 1
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 1
agrgtttgat cmtggctcag 20
<210> 2
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 2
ggttaccttg ttacgactt 19
<210> 3
<211> 1549
<212> DNA
<213> Bacillus megaterium
<400> 3
tcggagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcctaata catgcaagtc 60
gagcgaactg attagaagct tgcttctatg acgttagcgg cggacgggtg agtaacacgt 120
gggcaacctg cctgtaagac tgggataact tcgggaaacc gaagctaata ccggatagga 180
tcttctcctt catgggagat gattgaaaga tggtttcggc tatcacttac agatgggccc 240
gcggtgcatt agctagttgg tgaggtaacg gctcaccaag gcaacgatgc atagccgacc 300
tgagagggtg atcggccaca ctgggactga gacacggccc agactcctac gggaggcagc 360
agtagggaat cttccgcaat ggacgaaagt ctgacggagc aacgccgcgt gagtgatgaa 420
ggctttcggg tcgtaaaact ctgttgttag ggaagaacaa gtacgagagt aactgctcgt 480
accttgacgg tacctaacca gaaagccacg gctaactacg tgccagcagc cgcggtaata 540
cgtaggtggc aagcgttatc cggaattatt gggcgtaaag cgcgcgcagg cggtttctta 600
agtctgatgt gaaagcccac ggctcaaccg tggagggtca ttggaaactg gggaacttga 660
gtgcagaaga gaaaagcgga attccacgtg tagcggtgaa atgcgtagag atgtggagga 720
acaccagtgg cgaaggcggc tttttggtct gtaactgacg ctgaggcgcg aaagcgtggg 780
gagcaaacag gattagatac cctggtagtc cacgccgtaa acgatgagtg ctaagtgtta 840
gagggtttcc gccctttagt gctgcagcta acgcattaag cactccgcct ggggagtacg 900
gtcgcaagac tgaaactcaa aggaattgac gggggcccgc acaagcggtg gagcatgtgg 960
tttaattcga agcaacgcga agaaccttac caggtcttga catcctctga caactctaga 1020
gatagagcgt tccccttcgg gggacagagt gacaggtggt gcatggttgt cgtcagctcg 1080
tgtcgtgaga tgttgggtta agtcccgcaa cgagcgcaac ccttgatctt agttgccagc 1140
atttagttgg gcactctaag gtgactgccg gtgacaaacc ggaggaaggt ggggatgacg 1200
tcaaatcatc atgcccctta tgacctgggc tacacacgtg ctacaatgga tggtacaaag 1260
ggctgcaaga ccgcgaggtc aagccaatcc cataaaacca ttctcagttc ggattgtagg 1320
ctgcaactcg cctacatgaa gctggaatcg ctagtaatcg cggatcagca tgccgcggtg 1380
aatacgttcc cgggccttgt acacaccgcc cgtcacacca cgagagtttg taacacccga 1440
agtcggtgga gtaaccgtaa ggagctagcc gcctaaggtg ggacagatga ttggggtgaa 1500
gtcgtaacaa ggtagccgta tcggaaggtg cggctggatc acctccttt 1549
<210> 4
<211> 1567
<212> DNA
<213> Lactobacillus casei/paracasei
<400> 4
tatgagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcctaata catgcaagtc 60
gaacgagttc tcgttgatga tcggtgcttg caccgagatt caacatggaa cgagtggcgg 120
acgggtgagt aacacgtggg taacctgccc ttaagtgggg gataacattt ggaaacagat 180
gctaataccg catagatcca agaaccgcat ggttcttggc tgaaagatgg cgtaagctat 240
cgcttttgga tggacccgcg gcgtattagc tagttggtga ggtaacggct caccaaggcg 300
atgatacgta gccgaactga gaggttgatc ggccacattg ggactgagac acggcccaaa 360
ctcctacggg aggcagcagt agggaatctt ccacaatgga cgcaagtctg atggagcaac 420
gccgcgtgag tgaagaaggc tttcgggtcg taaaactctg ttgttggaga agaatggtcg 480
gcagagtaac tgttgtcggc gtgacggtat ccaaccagaa agccacggct aactacgtgc 540
cagcagccgc ggtaatacgt aggtggcaag cgttatccgg atttattggg cgtaaagcga 600
gcgcaggcgg ttttttaagt ctgatgtgaa agccctcggc ttaaccgagg aagcgcatcg 660
gaaactggga aacttgagtg cagaagagga cagtggaact ccatgtgtag cggtgaaatg 720
cgtagatata tggaagaaca ccagtggcga aggcggctgt ctggtctgta actgacgctg 780
aggctcgaaa gcatgggtag cgaacaggat tagataccct ggtagtccat gccgtaaacg 840
atgaatgcta ggtgttggag ggtttccgcc cttcagtgcc gcagctaacg cattaagcat 900
tccgcctggg gagtacgacc gcaaggttga aactcaaagg aattgacggg ggcccgcaca 960
agcggtggag catgtggttt aattcgaagc aacgcgaaga accttaccag gtcttgacat 1020
cttttgatca cctgagagat caggtttccc cttcgggggc aaaatgacag gtggtgcatg 1080
gttgtcgtca gctcgtgtcg tgagatgttg ggttaagtcc cgcaacgagc gcaaccctta 1140
tgactagttg ccagcattta gttgggcact ctagtaagac tgccggtgac aaaccggagg 1200
aaggtgggga tgacgtcaaa tcatcatgcc ccttatgacc tgggctacac acgtgctaca 1260
atggatggta caacgagttg cgagaccgcg aggtcaagct aatctcttaa agccattctc 1320
agttcggact gtaggctgca actcgcctac acgaagtcgg aatcgctagt aatcgcggat 1380
cagcacgccg cggtgaatac gttcccgggc cttgtacaca ccgcccgtca caccatgaga 1440
gtttgtaaca cccgaagccg gtggcgtaac ccttttaggg agcgagccgt ctaaggtggg 1500
acaaatgatt agggtgaagt cgtaacaagg tagccgtagg agaacctgcg gctggatcac 1560
ctccttt 1567
<210> 5
<211> 1510
<212> DNA
<213> Clostridium beijerinckii
<400> 5
tattgagagt ttgatcctgg ctcaggacga acgctggcgg cgtgcttaac acatgcaagt 60
cgagcgatga agttccttcg ggaatggatt agcggcggac gggtgagtaa cacgtgggta 120
acctgcctca tagaggggaa tagcctttcg aaaggaagat taataccgca taagattgta 180
gtgccgcatg gcatagcaat taaaggagta atccgctatg agatggaccc gcgtcgcatt 240
agctagttgg tgaggtaacg gctcaccaag gcgacgatgc gtagccgacc tgagagggtg 300
atcggccaca ttgggactga gacacggccc agactcctac gggaggcagc agtggggaat 360
attgcacaat gggggaaacc ctgatgcagc aacgccgcgt gagtgatgac ggtcttcgga 420
ttgtaaagct ctgtcttcag ggacgataat gacggtacct gaggaggaag ccacggctaa 480
ctacgtgcca gcagccgcgg taatacgtag gtggcaagcg ttgtccggat ttactgggcg 540
taaagggagc gtaggtggat atttaagtgg gatgtgaaat actcgggctt aacctgggtg 600
ctgcattcca aactggatat ctagagtgca ggagaggaaa gtagaattcc tagtgtagcg 660
gtgaaatgcg tagagattag gaagaatacc agtggcgaag gcgactttct ggactgtaac 720
tgacactgag gctcgaaagc gtggggagca aacaggatta gataccctgg tagtccacgc 780
cgtaaacgat gaatactagg tgtaggggtt gtcatgacct ctgtgccgcc gctaacgcat 840
taagtattcc gcctggggag tacggtcgca agattaaaac tcaaaggaat tgacgggggc 900
ccgcacaagc agcggagcat gtggtttaat tcgaagcaac gcgaagaacc ttacctagac 960
ttgacatctc ctgaattacc cttaatcggg gaagcccttc ggggcaggaa gacaggtggt 1020
gcatggttgt cgtcagctcg tgtcgtgaga tgttgggtta agtcccgcaa cgagcgcaac 1080
ccttattgtt agttgctacc atttagttga gcactctagc gagactgccc gggttaaccg 1140
ggaggaaggt ggggatgacg tcaaatcatc atgcccctta tgtctagggc tacacacgtg 1200
ctacaatggc tggtacagag agatgctaaa ccgtgaggtg gagccaaact ttaaaaccag 1260
tctcagttcg gattgtaggc tgaaactcgc ctacatgaag ctggagttgc tagtaatcgc 1320
gaatcagaat gtcgcggtga atacgttccc gggccttgta cacaccgccc gtcacaccat 1380
gagagttggc aatacccaaa gttcgtgagc taacgcgcaa gcggggcagc gacctaaggt 1440
agggtcagcg attggggtga agtcgtaaca aggtagccgt aggagaacct gcggctggat 1500
cacctccttt 1510
<210> 6
<211> 1485
<212> DNA
<213> Acetobacter pasteurianus
<400> 6
cctgagagtt tgatcctggc tcagagcgaa cgctggcggc atgcttaaca catgcaagtc 60
gcacgaaggt ttcggcctta gtggcggacg ggtgagtaac gcgtaggtat ctatccatgg 120
gtgggggata acactgggaa actggtgcta ataccgcatg acacctgagg gtcaaaggcg 180
caagtcgcct gtggaggagc ctgcgtttga ttagctagtt ggtggggtaa aggcctacca 240
aggcgatgat caatagctgg tttgagagga tgatcagcca cactgggact gagacacggc 300
ccagactcct acgggaggca gcagtgggga atattggaca atgggggcaa ccctgatcca 360
gcaatgccgc gtgtgtgaag aaggtcttcg gattgtaaag cactttcgac ggggacgatg 420
atgacggtac ccgtagaaga agccccggct aacttcgtgc cagcagccgc ggtaatacga 480
agggggctag cgttgctcgg aatgactggg cgtaaagggc gtgtaggcgg tttgtacagt 540
cagatgtgaa atccccgggc ttaacctggg agctgcattt gatacgtgca gactagagtg 600
tgagagaggg ttgtggaatt cccagtgtag aggtgaaatt cgtagatatt gggaagaaca 660
ccggtggcga aggcggcaac ctggctcatt actgacgctg aggcgcgaaa gcgtggggag 720
caaacaggat tagataccct ggtagtccac gctgtaaacg atgtgtgcta gatgttgggt 780
gacttagtca ttcagtgtcg cagttaacgc gttaagcaca ccgcctgggg agtacggccg 840
caaggttgaa actcaaagga attgacgggg gcccgcacaa gcggtggagc atgtggttta 900
attcgaagca acgcgcagaa ccttaccagg gcttgaatgt agaggctgca agcagagatg 960
tttgtttccc gcaagggacc tctaacacag gtgctgcatg gctgtcgtca gctcgtgtcg 1020
tgagatgttg ggttaagtcc cgcaacgagc gcaaccccta tctttagttg ccatcaggtt 1080
gggctgggca ctctagagag actgccggtg acaagccgga ggaaggtggg gatgacgtca 1140
agtcctcatg gcccttatgt cctgggctac acacgtgcta caatggcggt gacagtggga 1200
agctaggtgg tgacaccatg ctgatctcta aaagccgtct cagttcggat tgcactctgc 1260
aactcgagtg catgaaggtg gaatcgctag taatcgcgga tcagcatgcc gcggtgaata 1320
cgttcccggg ccttgtacac accgcccgtc acaccatggg agttggtttg accttaagcc 1380
ggtgagcgaa ccgcaaggac gcagccgacc acggtcgggt cagcgactgg ggtgaagtcg 1440
taacaaggta gccgtagggg aacctgcggc tggatcacct ccttt 1485
<210> 7
<211> 1571
<212> DNA
<213> Lactobacillus buchneri
<400> 7
atgagagttt gatcctggct caggacgaac gctggcggcg tgcctaatac atgcaagtcg 60
aacgcgtctc cgttaatgat tttaggtgct tgcacttgaa agatttaaca ttgagacgag 120
tggcgaactg gtgagtaaca cgtgggtaac ctgcccttga agtaggggat aacacttgga 180
aacaggtgct aataccgtat aacaaccaaa accacctggt tttggtttaa aagacggctt 240
cggctgtcac tttaggatgg acccgcggcg tattagcttg ttggtaaggt aacggcctac 300
caaggcgatg atacgtagcc gacctgagag ggtaatcggc cacattggga ctgagacacg 360
gcccaaactc ctacgggagg cagcagtagg gaatcttcca caatggacga aagtctgatg 420
gagcaacgcc gcgtgagtga tgaagggttt cggctcgtaa aactctgttg ttggagaaga 480
acaggtgtca gagtaactgt tgacatcttg acggtatcca accagaaagc cacggctaac 540
tacgtgccag cagccgcggt aatacgtagg tggcaagcgt tgtccggatt tattgggcgt 600
aaagcgagcg caggcggttt tttaggtctg atgtgaaagc cttcggctta accggagaag 660
tgcatcggaa accgggagac ttgagtgcag aagaggacag tggaactcca tgtgtagcgg 720
tgaaatgcgt agatatatgg aagaacacca gtggcgaagg cggctgtctg gtctgtaact 780
gacgctgagg ctcgaaagca tgggtagcga acaggattag ataccctggt agtccatgcc 840
gtaaacgatg agtgctaagt gttggagggt ttccgccctt cagtgctgca gctaacgcat 900
taagcactcc gcctggggag tacgaccgca aggttgaaac tcaaaggaat tgacgggggc 960
ccgcacaagc ggtggagcat gtggtttaat tcgatgctac gcgaagaacc ttaccaggtc 1020
ttgacatctt ctgccaactt aagagattag gcgttccctt cggggacaga atgacaggtg 1080
gtgcatggtt gtcgtcagct cgtgtcgtga gatgttgggt taagtcccgc aacgagcgca 1140
acccttattg ttagttgcca gcattcagtt gggcactcta gcaagactgc cggtgacaaa 1200
ccggaggaag gtggggatga cgtcaaatca tcatgcccct tatgacctgg gctacacacg 1260
tgctacaatg gacggtacaa cgagtcgcga aaccgcgagg tcaagctaat ctcttaaagc 1320
cgttctcagt tcggattgta ggctgcaact cgcctacatg aagttggaat cgctagtaat 1380
cgtggatcag catgccacgg tgaatacgtt cccgggcctt gtacacaccg cccgtcacac 1440
catgagagtt tgtaacaccc aaagccggtg aggtaacctt cggggaccag ccgtctaagg 1500
tggggcagat gattagggtg aagtcgtaac aaggtagccg taggagaacc tgcggctgga 1560
tcacctcctt t 1571
<210> 8
<211> 1548
<212> DNA
<213> Bacillus subtilis
<400> 8
atcggagagt ttgatcctgg ctcaggacga acgctggcgg cgtgcctaat acatgcaagt 60
cgagcggaca gatgggagct tgctccctga tgttagcggc ggacgggtga gtaacacgtg 120
ggtaacctgc ctgtaagact gggataactc cgggaaaccg gggctaatac cggatggttg 180
tttgaaccgc atggttcaaa cataaaaggt ggcttcggct accacttaca gatggacccg 240
cggcgcatta gctagttggt gaggtaacgg ctcaccaagg caacgatgcg tagccgacct 300
gagagggtga tcggccacac tgggactgag acacggccca gactcctacg ggaggcagca 360
gtagggaatc ttccgcaatg gacgaaagtc tgacggagca acgccgcgtg agtgatgaag 420
gttttcggat cgtaaagctc tgttgttagg gaagaacaag taccgttcga atagggcggt 480
accttgacgg tacctaacca gaaagccacg gctaactacg tgccagcagc cgcggtaata 540
cgtaggtggc aagcgttgtc cggaattatt gggcgtaaag ggctcgcagg cggtttctta 600
agtctgatgt gaaagccccc ggctcaaccg gggagggtca ttggaaactg gggaacttga 660
gtgcagaaga ggagagtgga attccacgtg tagcggtgaa atgcgtagag atgtggagga 720
acaccagtgg cgaaggcgac tctctggtct gtaactgacg ctgaggagcg aaagcgtggg 780
gagcgaacag gattagatac cctggtagtc cacgccgtaa acgatgagtg ctaagtgtta 840
gggggtttcc gccccttagt gctgcagcta acgcattaag cactccgcct ggggagtacg 900
gtcgcaagac tgaaactcaa aggaattgac gggggcccgc acaagcggtg gagcatgtgg 960
tttaattcga agcaacgcga agaaccttac caggtcttga catcctctga caatcctaga 1020
gataggacgt ccccttcggg ggcagagtga caggtggtgc atggttgtcg tcagctcgtg 1080
tcgtgagatg ttgggttaag tcccgcaacg agcgcaaccc ttgatcttag ttgccagcat 1140
tcagttgggc actctaaggt gactgccggt gacaaaccgg aggaaggtgg ggatgacgtc 1200
aaatcatcat gccccttatg acctgggcta cacacgtgct acaatggaca gaacaaaggg 1260
cagcgaaacc gcgaggttaa gccaatccca caaatctgtt ctcagttcgg atcgcagtct 1320
gcaactcgac tgcgtgaagc tggaatcgct agtaatcgcg gatcagcatg ccgcggtgaa 1380
tacgttcccg ggccttgtac acaccgcccg tcacaccacg agagtttgta acacccgaag 1440
tcggtgaggt aaccttttag gagccagccg ccgaaggtgg gacagatgat tggggtgaag 1500
tcgtaacaag gtagccgtat cggaaggtgc ggctggatca cctccttt 1548
<210> 9
<211> 1552
<212> DNA
<213> Paenibacillus cookii
<400> 9
cttggagagt ttgatcctgg ctcaggacga acgctggcgg cgtgcctaat acatgcaagt 60
cgagcggagt tgatggggag cttgctctcc tgagacttag cggcggacgg gtgagtaaca 120
cgtaggcaac ctgcccgtaa gaccgggata actaccggaa acggtagcta ataccggata 180
atttatcgct tcgcatggag cggtaatgaa agacggagca atctgtcact tacggatggg 240
cctgcggcgc attagctagt tggtgaggta acggctcacc aaggcgacga tgcgtagccg 300
acctgagagg gtgaacggcc acactgggac tgagacacgg cccagactcc tacgggaggc 360
agcagtaggg aatcttccgc aatgggcgaa agcctgacgg agcaacgccg cgtgagtgat 420
gaaggttttc ggatcgtaaa gctctgttgc cagggaagaa cgtcgggtag agtaactgct 480
atccgagtga cggtacctga gaagaaagcc ccggctaact acgtgccagc agccgcggta 540
atacgtaggg ggcaagcgtt gtccggaatt attgggcgta aagcgcgcgc aggcggtcac 600
ttaagtctgg tgtttaaggc tagggctcaa ctctagttcg cactggaaac tgggtgactt 660
gagtgcagaa gaggaaagtg gaattccacg tgtagcggtg aaatgcgtag agatgtggag 720
gaacaccagt ggcgaaggcg actttctggg ctgtaactga cgctgaggcg cgaaagcgtg 780
gggagcaaac aggattagat accctggtag tccacgccgt aaacgatgaa tgctaggtgt 840
taggggtttc gatacccttg gtgccgaagt taacacatta agcattccgc ctggggagta 900
cggtcgcaag actgaaactc aaaggaattg acggggaccc gcacaagcag tggagtatgt 960
ggtttaattc gaagcaacgc gaagaacctt accaggtctt gacatccctc tgaatcctct 1020
agagatagag gcggccttcg ggacagagga gacaggtggt gcatggttgt cgtcagctcg 1080
tgtcgtgaga tgttgggtta agtcccgcaa cgagcgcaac ccttgatttt agttgccagc 1140
acattaaggt gggcactcta gaatgactgc cggtgacaaa ccggaggaag gcggggatga 1200
cgtcaaatca tcatgcccct tatgacctgg gctacacacg tactacaatg gccagtacaa 1260
cgggaagcga agtcgcgaga cggagccaat cctatcaaag ctggtctcag ttcggattgc 1320
aggctgcaac ccgcctgcat gaagtcggaa ttgctagtaa tcgcggatca gcatgccgcg 1380
gtgaatacgt tcccgggtct tgtacacacc gcccgtcaca ccacgagagt ttacaacacc 1440
cgaagtcggt ggggtaaccg caaggagcca gccgccgaag gtggggtaga tgattggggt 1500
gaagtcgtaa caaggtagcc gtatcggaag gtgcggctgg atcacctcct tt 1552
<210> 10
<211> 1554
<212> DNA
<213> Lactobacillus vini
<400> 10
aatgagagtt tgatcctggc tcaggacgaa cgctggcggc gtgcctaata catgcaagtc 60
gaacgagact ttttatttga tgcttgcatc ttttaaaaag ttgagtggcg aacgggtgag 120
taacacgtgg gtaacctgcc ttaaagtggg ggataacact tggaaacagg tgctaatacc 180
gcataaccat caaaaccgcc tggttttgat gttaaagatg gttctgctat cgctttaaga 240
tggacccgcg gcgtattagc tagttggtga ggtaacggct taccaaggca atgatacgta 300
gccgaactga gaggttgatc ggccacattg ggactgagac acggcccaaa ctcctacggg 360
aggcagcagt agggaatctt tcacaatgga cgaaagtctg atggagcaac gccgcgtgag 420
tgaagaaggt tttcggatcg taaaactctg ttgtcagaga agaacgtgtg tgagagtaac 480
tgttcacgca gtgacggtat ctgaccagaa agtcacggct aactacgtgc cagcagccgc 540
ggtaatacgt aggtggcaag cgttgtccgg atttattggg cgtaaaggga acgcaggcgg 600
tcttttaagt ctgatgtgaa agccttcggc ttaaccgaag tcgggcattg gaaactggga 660
gacttgagtg cagaagagga gagtggaact ccatgtgtag cggtgaaatg cgtagatata 720
tggaagaaca ccagtggcga aagcggctct ctggtctgta actgacgctg aggttcgaaa 780
gcgtgggtag caaacaggat tagataccct ggtagtccac gccgtaaacg atgaatgcta 840
agtgttggag ggtttccgcc cttcagtgcc gcagctaacg cattaagcat tccgcctggg 900
gagtacgatc gcaagattga aactcaaagg aattgacggg ggcccgcaca agcggtggag 960
catgtggttt aattcgaagc aacgcgaaga accttaccag gtcttgacat cttttgctaa 1020
cctgagagat caggtgttcc cttcggggac aaaatgacag gtggtgcatg gttgtcgtca 1080
gctcgtgtcg tgagatgttg ggttaagtcc cgcaacgagc gcaaccctta ttgttagttg 1140
ccagcattta gttgggcact ctaacgagac tgccggtgac aaaccggagg aaggtgggga 1200
tgacgtcaaa tcatcatgcc ccttatgacc tgggctacac acgtgctaca atggacggta 1260
caacgagtcg caagaccgcg aggtcaagct aatctctgaa aaccgttctc agttcggatt 1320
gcaggctgca actcgcctgc atgaagtcgg aatcgctagt aatcgcggat cagcatgccg 1380
cggtgaatac gttcccgggc cttgtacaca ccgcccgtca caccatgaga gtttgtaaca 1440
cccaaagccg gtggggtaac ctttgggagc cagccgtcta aggtgggaca gatgattggg 1500
gtgaagtcgt aacaaggtag ccgtaggaga acctgcggct ggatcacctc cttt 1554
<210> 11
<211> 1549
<212> DNA
<213> Bacillus licheniformis
<400> 11
catggagagt ttgatcctgg ctcaggacga acgctggcgg cgtgcctaat acatgcaagt 60
cgagcggacc gacgggagct tgctccctta ggtcagcggc ggacgggtga gtaacacgtg 120
ggtaacctgc ctgtaagact gggataactc cgggaaaccg gggctaatac cggatgcttg 180
attgaaccgc atggttccaa tcataaaagg tggcttttag ctaccactta cagatggacc 240
cgcggcgcat tagctagttg gtgaggtaac ggctcaccaa ggcgacgatg cgtagccgac 300
ctgagagggt gatcggccac actgggactg agacacggcc cagactccta cgggaggcag 360
cagtagggaa tcttccgcaa tggacgaaag tctgacggag caacgccgcg tgagtgatga 420
aggttttcgg atcgtaaaac tctgttgtta gggaagaaca agtaccgttc gaatagggcg 480
gcaccttgac ggtacctaac cagaaagcca cggctaacta cgtgccagca gccgcggtaa 540
tacgtaggtg gcaagcgttg tccggaatta ttgggcgtaa agcgcgcgca ggcggtttct 600
taagtctgat gtgaaagccc ccggctcaac cggggagggt cattggaaac tggggaactt 660
gagtgcagaa gaggagagtg gaattccacg tgtagcggtg aaatgcgtag agatgtggag 720
gaacaccagt ggcgaaggcg actctctggt ctgtaactga cgctgaggcg cgaaagcgtg 780
gggagcgaac aggattagat accctggtag tccacgccgt aaacgatgag tgctaagtgt 840
tagagggttt ccgcccttta gtgctgcagc aaacgcatta agcactccgc ctggggagta 900
cggtcgcaag actgaaactc aaaggaattg acgggggccc gcacaagcgg tggagcatgt 960
ggtttaattc gaagcaacgc gaagaacctt accaggtctt gacatcctct gacaacccta 1020
gagatagggc ttccccttcg ggggcagagt gacaggtggt gcatggttgt cgtcagctcg 1080
tgtcgtgaga tgttgggtta agtcccgcaa cgagcgcaac ccttgatctt agttgccagc 1140
attcagttgg gcactctaag gtgactgccg gtgacaaacc ggaggaaggt ggggatgacg 1200
tcaaatcatc atgcccctta tgacctgggc tacacacgtg ctacaatggg cagaacaaag 1260
ggcagcgaag ccgcgaggct aagccaatcc cacaaatctg ttctcagttc ggatcgcagt 1320
ctgcaactcg actgcgtgaa gctggaatcg ctagtaatcg cggatcagca tgccgcggtg 1380
aatacgttcc cgggccttgt acacaccgcc cgtcacacca cgagagtttg taacacccga 1440
agtcggtgag gtaacctttt ggagccagcc gccgaaggtg ggacagatga ttggggtgaa 1500
gtcgtaacaa ggtagccgta tcggaaggtg cggctggatc acctccttt 1549
<210> 12
<211> 1553
<212> DNA
<213> Paenibacillus lautus
<400> 12
attggagagt ttgatcctgg ctcaggacga acgctggcgg cgtgcctaat acatgcaagt 60
cgagcggact tgatggagtg cttgcactcc tgaaggttag cggcggacgg gtgagtaaca 120
cgtaggcaac ctgccctcaa gactgggata actaccggaa acggtagcta ataccggata 180
atttattttg cagcattgtg aaataatgaa aggcggagca atctgtcact tgaggatggg 240
cctgcggcgc attagctagt tggtggggta acggcccacc aaggcgacga tgcgtagccg 300
acctgagagg gtgaacggcc acactgggac tgagacacgg cccagactcc tacgggaggc 360
agcagtaggg aatcttccgc aatgggcgaa agcctgacgg agcaacgccg cgtgagtgat 420
gaaggttttc ggatcgtaaa gctctgttgc caaggaagaa cgtcttctag agtaactgct 480
aggagagtga cggtacttga gaagaaagcc ccggctaact acgtgccagc agccgcggta 540
atacgtaggg ggcaagcgtt gtccggaatt attgggcgta aagcgcgcgc aggcggttct 600
ttaagtctgg tgtttaaacc cgaggctcaa cttcgggtcg cactggaaac tggggaactt 660
gagtgcagaa gaggagagtg gaattccacg tgtagcggtg aaatgcgtag atatgtggag 720
gaacaccagt ggcgaaggcg actctctggg ctgtaactga cgctgaggcg cgaaagcgtg 780
gggagcaaac aggattagat accctggtag tccacgccgt aaacgatgaa tgctaggtgt 840
taggggtttc gatacccttg gtgccgaagt taacacatta agcattccgc ctggggagta 900
cggtcgcaag actgaaactc aaaggaattg acggggaccc gcacaagcag tggagtatgt 960
ggtttaattc gaagcaacgc gaagaacctt accaagtctt gacatccctc tgaatcctct 1020
agagatagag gcggccttcg ggacagaggt gacaggtggt gcatggttgt cgtcagctcg 1080
tgtcgtgaga tgttgggtta agtcccgcaa cgagcgcaac ccttgatttt agttgccagc 1140
acttcgggtg ggcactctag aatgactgcc ggtgacaaac cggaggaagg cggggatgac 1200
gtcaaatcat catgcccctt atgacttggg ctacacacgt actacaatgg ctggtacaac 1260
gggaagcgaa gccgcgaggt ggagccaatc ctataaaagc cagtctcagt tcggattgca 1320
ggctgcaact cgcctgcatg aagtcggaat tgctagtaat cgcggatcag catgccgcgg 1380
tgaatacgtt cccgggtctt gtacacaccg cccgtcacac cacgagagtt tacaacaccc 1440
gaagtcggtg gggtaaccct taggggagcc agccgccgaa ggtggggtag atgattgggg 1500
tgaagtcgta acaaggtagc cgtatcggaa ggtgcggctg gatcacctcc ttt 1553
<210> 13
<211> 1561
<212> DNA
<213> Oceanobacillus oncorhynchi
<400> 13
ttatggagag tttgatcttg gctcaggacg aacgctggcg gcgtgcctaa tacatgcaag 60
tcgagcgcgg gaagcgaacg gaactcttcg gagggaagtt cgtggaacga gcggcggacg 120
ggtgagtaac acgtaggcaa cctgcctgta agactgggat aactcgcgga aacgcgagct 180
aataccggat aacactttct atcacctgat ggaaagttga aaggcggctt ttgctgtcac 240
ttacagatgg gcctgcggcg cattagctag ttggtgaggt aacggctcac caaggcgacg 300
atgcgtagcc gacctgagag ggtgatcggc cacactggga ctgagacacg gcccagactc 360
ctacgggagg cagcagtagg gaatcttccg caatggacga aagtctgacg gagcaacgcc 420
gcgtgagtga tgaaggtttt cggatcgtaa aactctgttg tcagggaaga acaagtacga 480
tagtaactga tcgtaccttg acggtacctg accagaaagc cacggctaac tacgtgccag 540
cagccgcggt aatacgtagg tggcaagcgt tgtccggaat tattgggcgt aaagcgctcg 600
caggcggttc tttaagtctg atgtgaaatc ttgcggctca accgcaaacg tgcattggaa 660
actggaggac ttgagtgcag aagaggagag tggaattcca cgtgtagcgg tgaaatgcgt 720
agagatgtgg aggaacacca gtggcgaagg cgactctctg gtctgtaact gacgctgagg 780
agcgaaagcg tggggagcga acaggattag ataccctggt agtccacgcc gtaaacgatg 840
agtgctaggt gttagggggt ttccgcccct tagtgctgaa gttaacgcat taagcactcc 900
gcctggggag tacggccgca aggctgaaac tcaaaagaat tgacggggac ccgcacaagc 960
ggtggagcat gtggtttaat tcgaagcaac gcgaagaacc ttaccaggtc ttgacatcct 1020
ttgaccgctc tagagataga gttttccctt cggggacaaa gtgacaggtg gtgcatggtt 1080
gtcgtcagct cgtgtcgtga gatgttgggt taagtcccgc aacgagcgca acccttaatc 1140
ttagttgcca gcatttagtt gggcactcta aggtgactgc cggtgacaaa ccggaggaag 1200
gtggggatga cgtcaaatca tcatgcccct tatgacctgg gctacacacg tgctacaatg 1260
gacggaacaa agggaagcga acccgcgagg tccagcaaat cccataaaac cgttctcagt 1320
tcggattgca ggctgcaact cgcctgcatg aagccggaat cgctagtaat cgcggatcag 1380
catgccgcgg tgaatacgtt cccgggtctt gtacacaccg cccgtcacac cacgagagtt 1440
cgtaacaccc gaagtcggtg aggtaacctt ttggagccag ccgccgaagg tgggacgaat 1500
gattggggtg aagtcgtaac aaggtagccg tatcggaagg tgcggctgga tcacctcctt 1560
t 1561
<210> 14
<211> 1548
<212> DNA
<213> Bacillus amyloliquefaciens
<400> 14
atcggagagt ttgatcctgg ctcaggacga acgctggcgg cgtgcctaat acatgcaagt 60
cgagcggaca gatgggagct tgctccctga tgttagcggc ggacgggtga gtaacacgtg 120
ggtaacctgc ctgtaagact gggataactc cgggaaaccg gggctaatac cggatggttg 180
tctgaaccgc atggttcaga cataaaaggt ggcttcggct accacttaca gatggacccg 240
cggcgcatta gctagttggt gaggtaacgg ctcaccaagg cgacgatgcg tagccgacct 300
gagagggtga tcggccacac tgggactgag acacggccca gactcctacg ggaggcagca 360
gtagggaatc ttccgcaatg gacgaaagtc tgacggagca acgccgcgtg agtgatgaag 420
gttttcggat cgtaaagctc tgttgttagg gaagaacaag tgccgttcaa atagggcggc 480
accttgacgg tacctaacca gaaagccacg gctaactacg tgccagcagc cgcggtaata 540
cgtaggtggc aagcgttgtc cggaattatt gggcgtaaag ggctcgcagg cggtttctta 600
agtctgatgt gaaagccccc ggctcaaccg gggagggtca ttggaaactg gggaacttga 660
gtgcagaaga ggagagtgga attccacgtg tagcggtgaa atgcgtagag atgtggagga 720
acaccagtgg cgaaggcgac tctctggtct gtaactgacg ctgaggagcg aaagcgtggg 780
gagcgaacag gattagatac cctggtagtc cacgccgtaa acgatgagtg ctaagtgtta 840
gggggtttcc gccccttagt gctgcagcta acgcattaag cactccgcct ggggagtacg 900
gtcgcaagac tgaaactcaa aggaattgac gggggcccgc acaagcggtg gagcatgtgg 960
tttaattcga agcaacgcga agaaccttac caggtcttga catcctctga caatcctaga 1020
gataggacgt ccccttcggg ggcagagtga caggtggtgc atggttgtcg tcagctcgtg 1080
tcgtgagatg ttgggttaag tcccgcaacg agcgcaaccc ttgatcttag ttgccagcat 1140
tcagttgggc actctaaggt gactgccggt gacaaaccgg aggaaggtgg ggatgacgtc 1200
aaatcatcat gccccttatg acctgggcta cacacgtgct acaatggaca gaacaaaggg 1260
cagcgaaacc gcgaggttaa gccaatccca caaatctgtt ctcagttcgg atcgcagtct 1320
gcaactcgac tgcgtgaagc tggaatcgct agtaatcgcg gatcagcatg ccgcggtgaa 1380
tacgttcccg ggccttgtac acaccgcccg tcacaccacg agagtttgta acacccgaag 1440
tcggtgaggt aacctttatg gagccagccg ccgaaggtgg gacagatgat tggggtgaag 1500
tcgtaacaag gtagccgtat cggaaggtgc ggctggatca cctccttt 1548
<210> 15
<211> 1547
<212> DNA
<213> Bacillus sp.
<400> 15
attggagagt ttgatcctgg ctcaggacga acgctggcgg cgtgcctaat acatgcaagt 60
cgagcgaatc tgagggagct tgctcccaaa gattagcggc ggacgggtga gtaacacgtg 120
ggcaacctgc ctgtaagact gggataactc cgggaaaccg gggctaatac cggataatat 180
ctatttatac atataattag attgaaagat ggttctgcta tcacttacag atgggcccgc 240
ggcgcattag ctagttggtg aggtaacggc tcaccaaggc gacgatgcgt agccgacctg 300
agagggtgat cggccacact gggactgaga cacggcccag actcctacgg gaggcagcag 360
tagggaatct tccgcaatgg acgaaagtct gacggagcaa cgccgcgtga gtgatgaagg 420
ttttcggatc gtaaaactct gttgttaggg aagaacaagt atcggagtaa ctgccggtac 480
cttgacggta cctaaccaga aagccacggc taactacgtg ccagcagccg cggtaatacg 540
taggtggcaa gcgttgtccg gaattattgg gcgtaaagcg cgcgcaggcg gttccttaag 600
tctgatgtga aagcccacgg ctcaaccgtg gagggtcatt ggaaactggg gaacttgagt 660
gcagaagagg aaagtggaat tccaagtgta gcggtgaaat gcgtagagat ttggaggaac 720
accagtggcg aaggcgactt tctggtctgt aactgacgct gaggcgcgaa agcgtgggga 780
gcaaacagga ttagataccc tggtagtcca cgccgtaaac gatgagtgct aagtgttaga 840
gggtttccgc cctttagtgc tgcagcaaac gcattaagca ctccgcctgg ggagtacgac 900
cgcaaggttg aaactcaaag gaattgacgg gggcccgcac aagcggtgga gcatgtggtt 960
taattcgaag caacgcgaag aaccttacca ggtcttgaca tcctctgaca atcctagaga 1020
taggactttc cccttcgggg gacagagtga caggtggtgc atggttgtcg tcagctcgtg 1080
tcgtgagatg ttgggttaag tcccgcaacg agcgcaaccc ttgatcttag ttgccagcat 1140
ttagttgggc actctaaggt gactgccggt gacaaaccgg aggaaggtgg ggatgacgtc 1200
aaatcatcat gccccttatg acctgggcta cacacgtgct acaatggatg gtacaaaggg 1260
ctgcaagacc gcgaggttta gccaatccca taaaaccatt ctcagttcgg attgtaggct 1320
gcaactcgcc tacatgaagc cggaatcgct agtaatcgcg gatcagcatg ccgcggtgaa 1380
tacgttcccg ggccttgtac acaccgcccg tcacaccacg agagtttgta acacccgaag 1440
tcggtggggt aaccttttgg agccagccgc ctaaggtggg acagatgatt ggggtgaagt 1500
cgtaacaagg tagccgtatc ggaaggtgcg gctggatcac ctccttt 1547
<210> 16
<211> 1533
<212> DNA
<213> Pseudomonas putida
<400> 16
ctgaagagtt tgatcatggc tcagattgaa cgctggcggc aggcctaaca catgcaagtc 60
gagcggatga gaagagcttg ctcttcgatt cagcggcgga cgggtgagta atgcctagga 120
atctgcctgg tagtggggga caacgtttcg aaaggaacgc taataccgca tacgtcctac 180
gggagaaagc aggggacctt cgggccttgc gctatcagat gagcctaggt cggattagct 240
agttggtgag gtaatggctc accaaggcga cgatccgtaa ctggtctgag aggatgatca 300
gtcacactgg aactgagaca cggtccagac tcctacggga ggcagcagtg gggaatattg 360
gacaatgggc gaaagcctga tccagccatg ccgcgtgtgt gaagaaggtc ttcggattgt 420
aaagcacttt aagttgggag gaagggcatt aacctaatac gttagtgttt tgacgttacc 480
gacagaataa gcaccggcta actctgtgcc agcagccgcg gtaatacaga gggtgcaagc 540
gttaatcgga attactgggc gtaaagcgcg cgtaggtggt ttgttaagtt ggatgtgaaa 600
gccccgggct caacctggga actgcatcca aaactggcaa gctagagtac ggtagagggt 660
ggtggaattt cctgtgtagc ggtgaaatgc gtagatatag gaaggaacac cagtggcgaa 720
ggcgaccacc tggactgata ctgacactga ggtgcgaaag cgtggggagc aaacaggatt 780
agataccctg gtagtccacg ccgtaaacga tgtcaactag ccgttggaat ccttgagatt 840
ttagtggcgc agctaacgca ttaagttgac cgcctgggga gtacggccgc aaggttaaaa 900
ctcaaatgaa ttgacggggg cccgcacaag cggtggagca tgtggtttaa ttcgaagcaa 960
cgcgaagaac cttaccaggc cttgacatgc agagaacttt ccagagatgg attggtgcct 1020
tcgggaactc tgacacaggt gctgcatggc tgtcgtcagc tcgtgtcgtg agatgttggg 1080
ttaagtcccg taacgagcgc aacccttgtc cttagttacc agcacgtaat ggtgggcact 1140
ctaaggagac tgccggtgac aaaccggagg aaggtgggga tgacgtcaag tcatcatggc 1200
ccttacggcc tgggctacac acgtgctaca atggtcggta cagagggttg ccaagccgcg 1260
aggtggagct aatctcacaa aaccgatcgt agtccggatc gcagtctgca actcgactgc 1320
gtgaagtcgg aatcgctagt aatcgcgaat cagaatgtcg cggtgaatac gttcccgggc 1380
cttgtacaca ccgcccgtca caccatggga gtgggttgca ccagaagtag ctagtctaac 1440
cttcgggagg acggttacca cggtgtgatt catgactggg gtgaagtcgt aacaaggtag 1500
ccgtagggga acctgcggct ggatcacctc ctt 1533
<210> 17
<211> 1533
<212> DNA
<213> Pseudomonas sp.
<400> 17
ctgaagagtt tgatcatggc tcagattgaa cgctggcggc aggcctaaca catgcaagtc 60
gagcggatga cgggagcttg ctccttgatt cagcggcgga cgggtgagta atgcctagga 120
atctgcctgg tagtggggga caacgtttcg aaaggaacgc taataccgca tacgtcctac 180
gggagaaagc aggggacctt cgggccttgc gctatcagat gagcctaggt cggattagct 240
agtaggtgag gtaatggctc acctaggcga cgatccgtaa ctggtctgag aggatgatca 300
gtcacactgg aactgagaca cggtccagac tcctacggga ggcagcagtg gggaatattg 360
gacaatgggc gaaagcctga tccagccatg ccgcgtgtgt gaagaaggtc ttcggattgt 420
aaagcacttt aagttgggag gaagggcagt aagctaatac cttgctgttt tgacgttacc 480
gacagaataa gcaccggcta actctgtgcc agcagccgcg gtaatacaga gggtgcaagc 540
gttaatcgga attactgggc gtaaagcgcg cgtaggtggt tcgttaagtt ggatgtgaaa 600
gccccgggct caacctggga actgcatcca aaactggcga gctagagtat ggtagagggt 660
ggtggaattt cctgtgtagc ggtgaaatgc gtagatatag gaaggaacac cagtggcgaa 720
ggcgaccacc tggactgata ctgacactga ggtgcgaaag cgtggggagc aaacaggatt 780
agataccctg gtagtccacg ccgtaaacga tgtcaactag ccgttggaat ccttgagatt 840
ttagtggcgc agctaacgca ttaagttgac cgcctgggga gtacggccgc aaggttaaaa 900
ctcaaatgaa ttgacggggg cccgcacaag cggtggagca tgtggtttaa ttcgaagcaa 960
cgcgaagaac cttaccaggc cttgacatgc agagaacttt ccagagatgg attggtgcct 1020
tcgggaactc tgacacaggt gctgcatggc tgtcgtcagc tcgtgtcgtg agatgttggg 1080
ttaagtcccg taacgagcgc aacccttgtc cttagttacc agcacgttat ggtgggcact 1140
ctaaggagac tgccggtgac aaaccggagg aaggtgggga tgacgtcaag tcatcatggc 1200
ccttacggcc tgggctacac acgtgctaca atggtcggta cagagggttg ccaagccgcg 1260
aggtggagct aatctcacaa aaccgatcgt agtccggatc gcagtctgca actcgactgc 1320
gtgaagtcgg aatcgctagt aatcgcaaat cagaatgttg cggtgaatac gttcccgggc 1380
cttgtacaca ccgcccgtca caccatggga gtgggttgca ccagaagtag ctagtctaac 1440
cttcgggggg acggttacca cggtgtgatt catgactggg gtgaagtcgt aacaaggtag 1500
ccgtagggga acctgcggct ggatcacctc ctt 1533
<210> 18
<211> 1523
<212> DNA
<213> Streptomyces griseus
<400> 18
acggagagtt tgatcctggc tcaggacgaa cgctggcggc gtgcttaaca catgcaagtc 60
gaacgatgaa gcctttcggg gtggattagt ggcgaacggg tgagtaacac gtgggcaatc 120
tgcccttcac tctgggacaa gccctggaaa cggggtctaa taccggataa cactctgtcc 180
cgcatgggac ggggttaaaa gctccggcgg tgaaggatga gcccgcggcc tatcagcttg 240
ttggtggggt aatggcctac caaggcgacg acgggtagcc ggcctgagag ggcgaccggc 300
cacactggga ctgagacacg gcccagactc ctacgggagg cagcagtggg gaatattgca 360
caatgggcga aagcctgatg cagcgacgcc gcgtgaggga tgacggcctt cgggttgtaa 420
acctctttca gcagggaaga agcgagagtg acggtacctg cagaagaagc gccggctaac 480
tacgtgccag cagccgcggt aatacgtagg gcgcaagcgt tgtccggaat tattgggcgt 540
aaagagctcg taggcggctt gtcacgtcgg atgtgaaagc ccggggctta accccgggtc 600
tgcattcgat acgggctagc tagagtgtgg taggggagat cggaattcct ggtgtagcgg 660
tgaaatgcgc agatatcagg aggaacaccg gtggcgaagg cggatctctg ggccattact 720
gacgctgagg agcgaaagcg tggggagcga acaggattag ataccctggt agtccacgcc 780
gtaaacgttg ggaactaggt gttggcgaca ttccacgtcg tcggtgccgc agctaacgca 840
ttaagttccc cgcctgggga gtacggccgc aaggctaaaa ctcaaaggaa ttgacggggg 900
cccgcacaag cagcggagca tgtggcttaa ttcgacgcaa cgcgaagaac cttaccaagg 960
cttgacatat accggaaagc atcagagatg gtgcccccct tgtggtcggt atacaggtgg 1020
tgcatggctg tcgtcagctc gtgtcgtgag atgttgggtt aagtcccgca acgagcgcaa 1080
cccttgttct gtgttgccag catgcccttc ggggtgatgg ggactcacag gagactgccg 1140
gggtcaactc ggaggaaggt ggggacgacg tcaagtcatc atgcccctta tgtcttgggc 1200
tgcacacgtg ctacaatggc cggtacaatg agctgcgatg ccgcgaggcg gagcgaatct 1260
caaaaagccg gtctcagttc ggattggggt ctgcaactcg accccatgaa gtcggagttg 1320
ctagtaatcg cagatcagca ttgctgcggt gaatacgttc ccgggccttg tacacaccgc 1380
ccgtcacgtc acgaaagtcg gtaacacccg aagccggtgg cccaacccct tgtgggaggg 1440
agctgtcgaa ggtgggactg gcgattggga cgaagtcgta acaaggtagc cgtaccggaa 1500
ggtgcggctg gatcacctcc ttt 1523
<210> 19
<211> 1553
<212> DNA
<213> Paenibacillus chibensis
<400> 19
cttggagagt ttgatcctgg ctcaggacga acgctggcgg cgtgcctaat acatgcaagt 60
cgagcggagt tgatgaggtg cttgcacctc tgatgcttag cggcggacgg gtgagtaaca 120
cgtaggtaac ctgcctgtaa gactgggata actaccggaa acggtagcta ataccggata 180
atttattttc tctcctgggg agataatgaa agacggagca atctgtcact tacagatggg 240
cctgcggcgc attagctagt tggtgaggta acggctcacc aaggcgacga tgcgtagccg 300
acctgagagg gtgaacggcc acactgggac tgagacacgg cccagactcc tacgggaggc 360
agcagtaggg aatcttccgc aatggacgaa agtctgacgg agcaacgccg cgtgagtgat 420
gaaggttttc ggatcgtaaa gctctgttgc cagggaagaa cgtccggtag agtaactgct 480
accggagtga cggtacctga gaagaaagcc ccggctaact acgtgccagc agccgcggta 540
atacgtaggg ggcaagcgtt gtccggaatt attgggcgta aagcgcgcgc aggcggtcac 600
ttaagtctgg tgtttaaggc caaggctcaa ccttggttcg cactggaaac tgggtgactt 660
gagtgcagaa gaggagagtg gaattccacg tgtagcggtg aaatgcgtag atatgtggag 720
gaacaccagt ggcgaaggcg actctctggg ctgtaactga cgctgaggcg cgaaagcgtg 780
gggagcaaac aggattagat accctggtag tccacgccgt aaacgatgaa tgctaggtgt 840
taggggtttc gatacccttg gtgccgaagt taacacatta agcattccgc ctggggagta 900
cggtcgcaag actgaaactc aaaggaattg acggggaccc gcacaagcag tggagtatgt 960
ggtttaattc gaagcaacgc gaagaacctt accaagtctt gacatccctc tgaatcctct 1020
agagatagag gcggccttcg ggacagaggt gacaggtggt gcatggttgt cgtcagctcg 1080
tgtcgtgaga tgttgggtta agtcccgcaa cgagcgcaac ccttgatttt agttgccagc 1140
atttcggatg ggcactctag aatgactgcc ggtgacaaac cggaggaagg cggggatgac 1200
gtcaaatcat catgcccctt atgacttggg ctacacacgt actacaatgg ccagtacaac 1260
gggaagcgaa atcgcgagat ggagccaatc ctatcaaagc tggtctcagt tcggattgca 1320
ggctgcaacc cgcctgcatg aagtcggaat tgctagtaat cgcggatcag catgccgcgg 1380
tgaatacgtt cccgggtctt gtacacaccg cccgtcacac cacgagagtt tacaacaccc 1440
gaagtcggtg gggtaacccg caagggagcc agccgccgaa ggtggggtag atgattgggg 1500
tgaagtcgta acaaggtagc cgtatcggaa ggtgcggctg gatcacctcc ttt 1553
<210> 20
<211> 1550
<212> DNA
<213> Bacillus flexus
<400> 20
tcggagagtt tgatcctggc tcaggatgaa cgctggcggc gtgcctaata catgcaagtc 60
gagcgaactg attagaagct tgcttctatg acgttagcgg cggacgggtg agtaacacgt 120
gggcaacctg cctgtaagac tgggataact ccgggaaacc ggagctaata ccggataaca 180
ttttctcttg cataagagaa aattgaaaga tggtttcggc tatcacttac agatgggccc 240
gcggtgcatt agctagttgg tgaggtaacg gctcaccaag gcaacgatgc atagccgacc 300
tgagagggtg atcggccaca ctgggactga gacacggccc agactcctac gggaggcagc 360
agtagggaat cttccgcaat ggacgaaagt ctgacggagc aacgccgcgt gagtgatgaa 420
ggctttcggg tcgtaaaact ctgttgttag ggaagaacaa gtacaagagt aactgcttgt 480
accttgacgg tacctaacca gaaagccacg gctaactacg tgccagcagc cgcggtaata 540
cgtaggtggc aagcgttatc cggaattatt gggcgtaaag cgcgcgcagg cggtttctta 600
agtctgatgt gaaagcccac ggctcaaccg tggagggtca ttggaaactg gggaacttga 660
gtgcagaaga gaaaagcgga attccacgtg tagcggtgaa atgcgtagag atgtggagga 720
acaccagtgg cgaaggcggc tttttggtct gtaactgacg ctgaggcgcg aaagcgtggg 780
gagcaaacag gattagatac cctggtagtc cacgccgtaa acgatgagtg ctaagtgtta 840
gagggtttcc gccctttagt gctgcagcta acgcattaag cactccgcct ggggagtacg 900
gtcgcaagac tgaaactcaa aggaattgac gggggcccgc acaagcggtg gagcatgtgg 960
tttaattcga agcaacgcga agaaccttac caggtcttga catcctctga caactctaga 1020
gatagagcgt tccccttcgg gggacagagt gacaggtggt gcatggttgt cgtcagctcg 1080
tgtcgtgaga tgttgggtta agtcccgcaa cgagcgcaac ccttgatctt agttgccagc 1140
atttagttgg gcactctaag gtgactgccg gtgacaaacc ggaggaaggt ggggatgacg 1200
tcaaatcatc atgcccctta tgacctgggc tacacacgtg ctacaatgga tggtacaaag 1260
ggctgcaaga ccgcgaggtc aagccaatcc cataaaacca ttctcagttc ggattgtagg 1320
ctgcaactcg cctacatgaa gctggaatcg ctagtaatcg cggatcagca tgccgcggtg 1380
aatacgttcc cgggccttgt acacaccgcc cgtcacacca cgagagtttg taacacccga 1440
agtcggtggg gtaaccttta tggagccagc cgcctaaggt gggacagatg attggggtga 1500
agtcgtaaca aggtagccgt atcggaaggt gcggctggat cacctccttt 1550
<210> 21
<211> 1505
<212> DNA
<213> Clostridium pasteurianum
<400> 21
aattgagagt ttgatcctgg ctcaggacga acgctggcgg cgtgcttaac acatgcaagt 60
cgagcgagaa accttcgggt ttctagcggc ggacgggtga gtaacacgtg ggtaacctgc 120
ctcaaagagg ggaatagcct cccgaaaggg agattaatac cgcataatat tacagcttcg 180
catgaagcag taattaaagg agtaatccgc tttgagatgg acccgcggcg cattagctag 240
ttggagaggt aacggctcac caaggcgacg atgcgtagcc gacctgagag ggtgatcggc 300
cacattggaa ctgagacacg gtccagactc ctacgggagg cagcagtggg gaatattgca 360
caatgggcga aagcctgatg cagcaacgcc gcgtgagtga tgacggtctt cggattgtaa 420
agctctgtct tttgggacga taatgacggt accaaaggag gaagccacgg ctaactacgt 480
gccagcagcc gcggtaatac gtaggtggca agcgttgtcc ggatttactg ggcgtaaagg 540
atgtgtaggc ggatacttaa gtgagatgtg aaagccccgg gcttaacttg gggactgcat 600
ttcaaactgg gtgtctagag tgcaggagag gaaagcggaa ttcctagtgt agcggtgaaa 660
tgcgtagaga ttaggaagaa catcagtggc gaaggcggct ttctggactg taactgacgc 720
tgaggcatga aagcgtgggg agcaaacagg attagatacc ctggtagtcc acgccgtaaa 780
cgatgagtac taggtgtagg aggtatcgac tccttctgtg ccgcagtaaa cacaataagt 840
actccgcctg ggaagtacgg tcgcaagatt aaaactcaaa ggaattgacg ggggcccgca 900
caagcagcgg agcatgtggt ttaattcgaa gcaacgcgaa gaaccttacc tagacttgac 960
atctcctgaa tagcgtagag atacgtgaag cccttcgggg caggaagaca ggtggtgcat 1020
ggttgtcgtc agctcgtgtc gtgagatgtt gggttaagtc ccgcaacgag cgcaaccctt 1080
atcattagtt gctaccatta agttgagcac tctagtgaga ctgcccgggt taaccgggag 1140
gaaggcgggg atgacgtcaa atcatcatgc cccttatgtc tagggctaca cacgtgctac 1200
aatggtgaga acaacgagat gcaataccgc gaggtggagc caaacttgaa aactcatccc 1260
agttcggatt gtaggctgaa attcgcctac atgaagttgg agttgctagt aatcgcgaat 1320
cagaatgtcg cggtgaatac gttcccgggc cttgtacaca ccgcccgtca caccatgaga 1380
gctggtaaca cccgaagtcc gtgaggtaac ctttatggag ccagcggccg aaggtgggat 1440
tagtgattgg ggtgaagtcg taacaaggta gccgtaggag aacctgcggc tggatcacct 1500
ccttt 1505
<210> 22
<211> 1532
<212> DNA
<213> Azotobacter vinelandii
<400> 22
ctgaagagtt tgatcatggc tcagattgaa cgctggcggc aggcctaaca catgcaagtc 60
gagcggcagc gggaccttcg ggttgccggc gagcggcgga cgggtgagta atgcctagga 120
atctgcctgt tagtggggga taacgcgggg aaactcgcgc taataccgca tacgtcctac 180
gggagaaagt gggggacctt cgggcctcac gctaacagat gagcctaggt cggattagct 240
ggttggtggg gtaacggccc accaaggcga cgatccgtaa ctggtctgag aggatgatca 300
gtcacactgg aactgagaca cggtccagac tcctacggga ggcagcagtg gggaatattg 360
gacaatgggc gaaagcctga tccagccatg ccgcgtgtgt gaagaaggtc ttcggattgt 420
aaagcacttt aagtcgggag gaagggctgt aggcgaatac cctgcagttt tgacgttacc 480
gacagaataa gcaccggcta acttcgtgcc agcagccgcg gtaatacgaa gggtgcaagc 540
gttaatcgga attactgggc gtaaagcgcg cgtaggtggt ttggtaagtt ggatgtgaaa 600
gccccgggct caacctggga actgcatcca aaactgccag gctagagtac ggtagagggt 660
ggtggaattt cctgtgtagc ggtgaaatgc gtagatatag gaaggaacac cagtggcgaa 720
ggcgaccacc tggactgata ctgacactga ggtgcgaaag cgtggggagc aaacaggatt 780
agataccctg gtagtccacg ccgtaaacga tgtcgactag ccgttgggct ccttgagagc 840
ttagtggcgc agctaacgca ttaagtcgac cgcctgggga gtacggccgc aaggttaaaa 900
ctcaaatgaa ttgacggggg cccgcacaag cggtggagca tgtggtttaa ttcgaagcaa 960
cgcgaagaac cttacctggc cttgacatcc tgcgaactgg gtagagatac ccgggtgcct 1020
tcgggaacgc agagacaggt gctgcatggc tgtcgtcagc tcgtgtcgtg agatgttggg 1080
ttaagtcccg taacgagcgc aacccttgtc cttagttacc agcacctcgg gtgggcactc 1140
taaggagact gccggtgaca aaccggagga aggtggggat gacgtcaagt catcatggcc 1200
cttacggcca gggctacaca cgtgctacaa tggtcggtac agagggttgc caagccgcga 1260
ggcggagcta atcccagaaa accgatcgta gtccggatcg cagtctgcaa ctcgactgcg 1320
tgaagtcgga atcgctagta atcgcgaatc agaatgtcgc ggtgaatacg ttcccgggcc 1380
ttgtacacac cgcccgtcac accatgggag tgggttgctc cagaagtagc tagtctaacc 1440
ctcgggagga cggttaccac ggagtgattc atgactgggg tgaagtcgta acaaggtagc 1500
cgtaggggaa cctgcggctg gatcacctcc tt 1532
<210> 23
<211> 1566
<212> DNA
<213> Virgibacillus halophilus
<400> 23
ttttggagag tttgatcttg gctcaggacg aacgctggcg gcgtgcctaa tacatgcaag 60
tcgagcgcgg gaagcaggat gatcctcatc tgaggtgatt cctgtggaac gagcggcgga 120
cgggtgagta acacgtgggc aacctgcctg taagatcggg ataactcgtg gaaacgcgag 180
ctaataccgg atgatacttt tcatcgcatg gtgagaagtt gaaagatggc tttaagctat 240
cacttacaga tgggcccgcg gcgcattagc tagttggtgg ggtaacggcc taccaaggca 300
acgatgcgta gccgacctga gagggtgatc ggccacactg ggactgagac acggcccaga 360
ctcctacggg aggcagcagt agggaatctt ccgcaatgga cgaaagtctg acggagcaac 420
gccgcgtgag tgatgaaggt tttcggatcg taaaactctg ttgtcaggga agaacaagtg 480
ccgtttgaat aaggcggcac cttgacggta cctgaccaga aagccccggc taactacgtg 540
ccagcagccg cggtaatacg tagggggcaa gcgttgtccg gaattattgg gcgtaaagcg 600
cgcgcaggcg gtcttttaag tctgatgtga aagcccacgg cttaaccgtg gagggtcatt 660
ggaaactgga ggacttgagt gcagaagagg agagtggaat tccatgtgta gcggtgaaat 720
gcgtagagat atggaggaac accagtggcg aaggcgactc tctggtctgc aactgacgct 780
gaggcgcgaa agcgtgggta gcgaacagga ttagataccc tggtagtcca cgccgtaaac 840
gatgagtgct aggtgttagg gggtttccgc cccttagtgc tgaagttaac gcattaagca 900
ctccgcctgg ggagtacggc cgcaaggctg aaactcaaaa gaattgacgg gggcccgcac 960
aagcggtgga gcatgtggtt taattcgaag caacgcgaag aaccttacca ggtcttgaca 1020
tcctctgaca gccttagaga taaggtgttc ccttcgggga cagagtgaca ggtggtgcat 1080
ggttgtcgtc agctcgtgtc gtgagatgtt gggttaagtc ccgcaacgag cgcaaccctt 1140
gagattagtt gccagcatta agttgggcac tctaatctga ctgccggtga caaaccggag 1200
gaaggtgggg atgacgtcaa atcatcatgc cccttatgac ctgggctaca cacgtgctac 1260
aatggatggt acagagggaa gcgaagccgc gaggtgaagc aaatcccaca aaaccattct 1320
cagttcggat tgcaggctgc aactcgcctg catgaagccg gaatcgctag taatcgcgga 1380
tcagcatgcc gcggtgaata cgttcccggg ccttgtacac accgcccgtc acaccacgag 1440
agttggtaac acccgaagtc ggtgaggtaa cctttttgga gccagccgcc gaaggtggga 1500
cgaatgattg gggtgaagtc gtaacaaggt agccgtatcg gaaggtgcgg ctggatcacc 1560
tccttt 1566
<210> 24
<211> 1561
<212> DNA
<213> Lactobacillus delbrueckii
<400> 24
attgagagtt tgatcctggc tcaggacgaa cgctggcggc gtgcctaata catgcaagtc 60
gagcgagctg aattcaaaga tcccttcggg gtgatttgtt ggacgctagc ggcggatggg 120
tgagtaacac gtgggcaatc tgccctaaag actgggatac cacttggaaa caggtgctaa 180
taccggataa caacatgaat cgcatgattc aagtttgaaa ggcggcgcaa gctgtcactt 240
taggatgagc ccgcggcgca ttagctagtt ggtggggtaa aggcctacca aggcaatgat 300
gcgtagccga gttgagagac tgatcggcca cattgggact gagacacggc ccaaactcct 360
acgggaggca gcagtaggga atcttccaca atggacgcaa gtctgatgga gcaacgccgc 420
gtgagtgaag aaggtcttcg gatcgtaaag ctctgttgtt ggtgaagaag gatagaggca 480
gtaactggtc tttatttgac ggtaatcaac cagaaagtca cggctaacta cgtgccagca 540
gccgcggtaa tacgtaggtg gcaagcgttg tccggattta ttgggcgtaa agcgagcgca 600
ggcggaatga taagtctgat gtgaaagccc acggcttaac cgtggaactg catcggaaac 660
tgtcattctt gagtgcagaa gaggagagtg gaactccatg tgtagcggtg gaatgcgtag 720
atatatggaa gaacaccagt ggcgaaggcg gctctctggt ctgcaactga cgctgaggct 780
cgaaagcatg ggtagcgaac aggattagat accctggtag tccatgccgt aaacgatgag 840
cgctaggtgt tggggacttt ccggttctca gtgccgcagc aaacgcgtta agcgctccgc 900
ctggggagta cgaccgcaag gttgaaactc aaaggaattg acgggggccc gcacaagcgg 960
tggagcatgt ggtttaattc gaagcaacgc gaagaacctt accaggtctt gacatcctgc 1020
gctacaccta gagataggtg gttcccttcg gggacgcaga gacaggtggt gcatggctgt 1080
cgtcagctcg tgtcgtgaga tgttgggtta agtcccgcaa cgagcgcaac ccttgtcttt 1140
agttgccatc attaagttgg gcactctaaa gagactgccg gtgacaaacc ggaggaaggt 1200
ggggatgacg tcaagtcatc atgcccctta tgacctgggc tacacacgtg ctacaatggg 1260
cagtacaacg agaagcgaac ccgcgagggt aagcggatct cttaaagctg ctctcagttc 1320
ggactgcagg ctgcaactcg cctgcacgaa gctggaatcg ctagtaatcg cggatcagca 1380
cgccgcggtg aatacgttcc cgggccttgt acacaccgcc cgtcacacca tggaagtctg 1440
caatgcccaa agtcggtgag ataaccttta taggagtcag ccgcctaagg cagggcagat 1500
gactggggtg aagtcgtaac aaggtagccg taggagaacc tgcggctgga tcacctcctt 1560
t 1561
<210> 25
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 25
caagtcgcac gaaggtttc 19
<210> 26
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 26
cggggatttc acatctgact 20
<210> 27
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 27
gggtcaagag cttcacctac 20
<210> 28
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 28
cgatgtctgc cagggaatg 19
<210> 29
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 29
tgcgcttatg aatggaggag 20
<210> 30
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 30
ctttatcagg cctggtaccg 20
<210> 31
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 31
tctcttgcat aagagaaaat tgaaa 25
<210> 32
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 32
ctacgcattt caccgctaca 20
<210> 33
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 33
ggagcttgct cccttaggtc 20
<210> 34
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 34
ctcaagttcc ccagtttcca 20
<210> 35
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 35
ccggatagga tcttctcctt c 21
<210> 36
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 36
ctacgcattt caccgctaca 20
<210> 37
<211> 29
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 37
tttatacata taattagatt gaaagatgg 29
<210> 38
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 38
ctacgcattt caccgctaca 20
<210> 39
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 39
gatctttctt gggggatggg 20
<210> 40
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 40
ccgaacccaa cagtccaata 20
<210> 41
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 41
gatgaagctc cttcgggagt 20
<210> 42
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 42
aatgcagcac ccaggttaag 20
<210> 43
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 43
caagtcgagc gagaaacctt 20
<210> 44
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 44
gaaatgcagt ccccaggtta 20
<210> 45
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 45
ggtgcttgca cttgaaagat t 21
<210> 46
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 46
ctcgctttac gcccaataaa 20
<210> 47
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 47
cgagcgagct gaattcaaag 20
<210> 48
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 48
ctcgctttac gcccaataaa 20
<210> 49
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 49
ctcgttgatg atcggtgct 19
<210> 50
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 50
taaatccgga taacgcttgc 20
<210> 51
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 51
accgcctggt tttgatgtta 20
<210> 52
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 52
catttcaccg ctacacatgg 20
<210> 53
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 53
ggaactcttc ggagggaagt 20
<210> 54
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 54
cagtttccaa tgcacgtttg 20
<210> 55
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 55
tgcagcattg tgaaataatg aa 22
<210> 56
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 56
ctacgcattt caccgctaca 20
<210> 57
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 57
ccggataatt tattttctct cctg 24
<210> 58
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 58
ctacgcattt caccgctaca 20
<210> 59
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 59
atttatcgct tcgcatggag 20
<210> 60
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 60
ctacgcattt caccgctaca 20
<210> 61
<211> 18
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 61
cgggagcttg ctccttga 18
<210> 62
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 62
ctctagctcg ccagttttgg 20
<210> 63
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 63
aagtcgagcg gatgagaaga 20
<210> 64
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 64
cgctttacgc ccagtaattc 20
<210> 65
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 65
gtcgaacgat gaagcctttc 20
<210> 66
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 66
aggaattccg atctccccta 20
<210> 67
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 67
gtgccagcag ccgcggtaa 19
<210> 68
<211> 27
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 68
tggactacca gggtatctaa tcctgtt 27
<210> 69
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 69
cctcatctga ggtgattcct g 21
<210> 70
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 70
tcctccagtt tccaatgacc 20
<210> 71
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 71
tcgagcgaaa cagaagtgaa 20
<210> 72
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide probe
<400> 72
tctgtgatga atgtgatgcg ga 22
<210> 73
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 73
gctgaacttt cacacgatgc 20
<210> 74
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 74
acgcaggcga tttatcatca 20
<210> 75
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide probe
<400> 75
tttgctttcc ggtggctcat 20
<210> 76
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 76
agcccatatc aaccagcatc 20
<210> 77
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 77
gctgaaggag ggacactttt 20
<210> 78
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide probe
<400> 78
agagtttgaa cgtgttggtg gt 22
<210> 79
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 79
tcagatcacg gtttgttgct 20
<210> 80
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 80
aacggttaac aatcagccca 20
<210> 81
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide probe
<400> 81
tcttaccgga aaagaattcg cca 23
<210> 82
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 82
cgcaagacaa gcagttcaag 20
<210> 83
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 83
caaccaactg gatcaaggga 20
<210> 84
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide probe
<400> 84
acctgctgaa gcagcgattt 20
<210> 85
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 85
aatcataccg atcagtgccg 20
<210> 86
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 86
tgctgaacgg aaaacatcct 20
<210> 87
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide probe
<400> 87
aaaatcggtg cggaaggtcc 20
<210> 88
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 88
tgcaactaca cttaccgcaa 20
<210> 89
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 89
tgcgcttatg aatggaggag 20
<210> 90
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide probe
<400> 90
aaaagggccg atcacatggg 20
<210> 91
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 91
ctgtaatccg gtccgtacac 20
<210> 92
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 92
atatcctgcg ctggtacaac 20
<210> 93
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide probe
<400> 93
cggcagactt gaagctcgag 20
<210> 94
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 94
tcttgtacat ggaagccgtg 20
<210> 95
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 95
cgactaacct gatcgcactt 20
<210> 96
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide probe
<400> 96
tgaagctcag atttcacggc t 21
<210> 97
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 97
attacgccga ttccttctgg 20
<210> 98
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 98
tgacattcca ttcatccggg 20
<210> 99
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide probe
<400> 99
aaatggcgga gatcacgtat ca 22
<210> 100
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 100
atcccagcca aatttccaca 20
<210> 101
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 101
gcaaaacaaa caggctccaa 20
<210> 102
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide probe
<400> 102
aaatcagcct ctggcttgcc 20
<210> 103
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 103
ctgaccggga tagttggttc 20
<210> 104
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 104
ctggatatcc cgcatttggt 20
<210> 105
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide probe
<400> 105
ctgtatgccg ctttgacgga 20
<210> 106
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 106
gcgaggaatc atgtagcctt 20
<210> 107
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 107
aggttccgat gtagtgcttg 20
<210> 108
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide probe
<400> 108
acatacaacg cacaccgaga a 21
<210> 109
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 109
atttcctgca accagagctt 20
<210> 110
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 110
tccgaagctg ctgaaatctt 20
<210> 111
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide probe
<400> 111
acctgaccgt ggtggagaaa 20
<210> 112
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 112
ttgaaagtaa atcgcgcgtc 20
<210> 113
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 113
aatcatcaca gatgcggagg 20
<210> 114
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide probe
<400> 114
attgtgccat ccggctatgg 20
<210> 115
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 115
gtgccgagat gaagaagtga 20
<210> 116
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 116
gctgacctat gtgaagtccc 20
<210> 117
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide probe
<400> 117
agatcgatgg cgtgttggtg 20
<210> 118
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 118
tgataaagat ggacgccgac 20
<210> 119
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 119
ctgggactac atgaagcagg 20
<210> 120
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide probe
<400> 120
tggacgccga gatcctctac 20
<210> 121
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 121
taggtcttct ggagcgactt 20
<210> 122
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 122
tgggcttggt gtatgtgttt 20
<210> 123
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide probe
<400> 123
atggcacaaa gctacggctt 20
<210> 124
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 124
gaaccatgag cccgtaatga 20
<210> 125
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 125
ggatgtacag aaaattgcag c 21
<210> 126
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide probe
<400> 126
agcttattgg agaggttaca actgt 25
<210> 127
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 127
aggagttgat atcaggtatg gt 22
<210> 128
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 128
gcatattgcc tttggtgtgg 20
<210> 129
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide probe
<400> 129
ccccatgggg gtaaccattg 20
<210> 130
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 130
tccatcctgg ggttgttttc 20
<210> 131
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 131
gctatatggg ctccaaagca 20
<210> 132
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide probe
<400> 132
aaaagccgat gtagtactcg ct 22
<210> 133
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 133
ttccgaaaac agacagcctt 20
<210> 134
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 134
accagtgaag aactggaagc 20
<210> 135
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide probe
<400> 135
acgcgaagac aagatctgcc 20
<210> 136
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic oligonucleotide primer
<400> 136
tcaccgttca aagtccagtc 20
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| ДНК-СОДЕРЖАЩИЕ ПОЛИНУКЛЕОТИДЫ И РУКОВОДЯЩИЕ ПОСЛЕДОВАТЕЛЬНОСТИ ДЛЯ СИСТЕМ CRISPR ТИПА V И СПОСОБЫ ИХ ПОЛУЧЕНИЯ И ПРИМЕНЕНИЯ | 2021 |
|
RU2832314C1 |
| СПОСОБ УСТАНОВЛЕНИЯ ПОДЛИННОСТИ МОЛОЧНЫХ ПРОДУКТОВ | 2010 |
|
RU2567658C2 |
| КОНСОРЦИУМЫ МИКРООРГАНИЗМОВ | 2016 |
|
RU2739079C2 |
| СПОСОБ ОТБОРА МИКРООРГАНИЗМОВ ДЛЯ ОБЪЕДИНЕНИЯ В КОМПОЗИЦИЮ С НОСИТЕЛЯМИ | 2019 |
|
RU2793460C2 |
| АНТИСМЫСЛОВЫЕ ОЛИГОНУКЛЕОТИДЫ К АЛЬФА-СИНУКЛЕИНУ И ИХ ПРИМЕНЕНИЯ | 2019 |
|
RU2773197C2 |
| СИСТЕМЫ CRISPR-CAS И СПОСОБЫ ИЗМЕНЕНИЯ ЭКСПРЕССИИ ПРОДУКТОВ ГЕНОВ | 2013 |
|
RU2796273C2 |
| МАНИПУЛЯЦИЯ МЕТАБОЛИЗМОМ ТРИПТАМИНОВ | 2018 |
|
RU2794244C2 |
| КОМПОЗИЦИИ ОЛИГОНУКЛЕОТИДОВ И СПОСОБЫ С НИМИ | 2016 |
|
RU2830607C2 |
| ШТАММ KBL396 LACTOBACILLUS PLANTARUM И ЕГО ПРИМЕНЕНИЕ | 2019 |
|
RU2764639C1 |
| ПЕЧЕНЬ-СПЕЦИФИЧНЫЕ КОНСТРУКЦИИ, ЭКСПРЕССИОННЫЕ КАССЕТЫ ФАКТОРА VIII И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2016 |
|
RU2774874C2 |
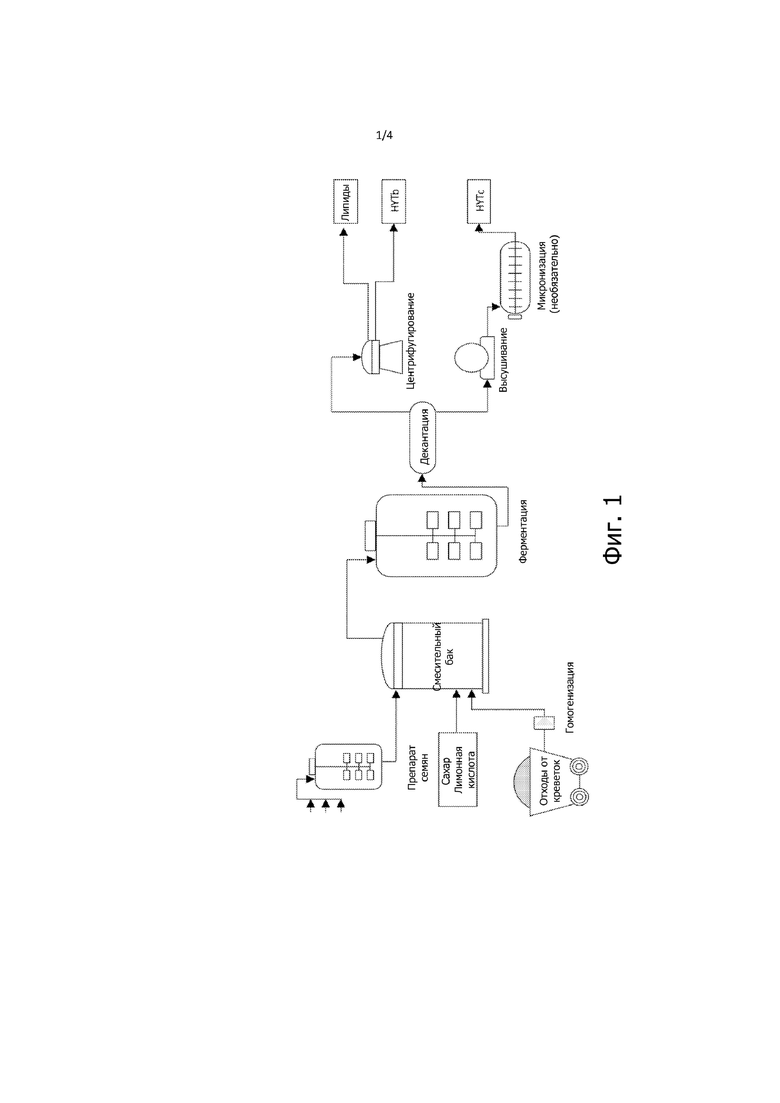
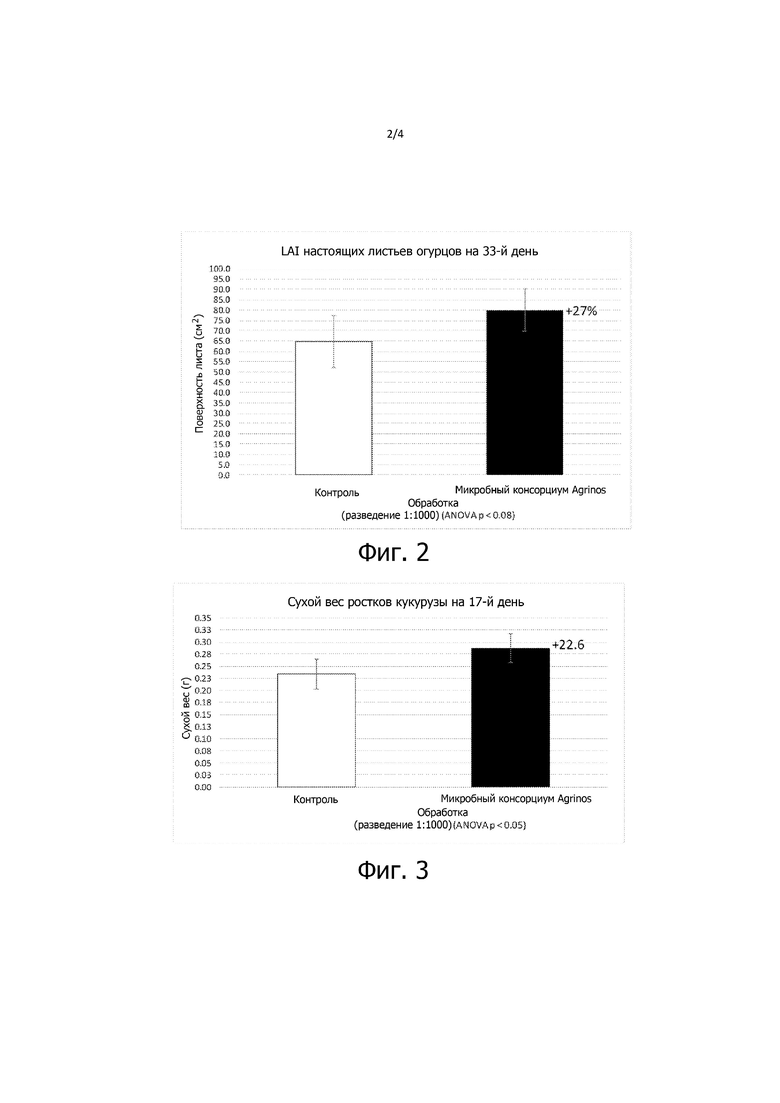
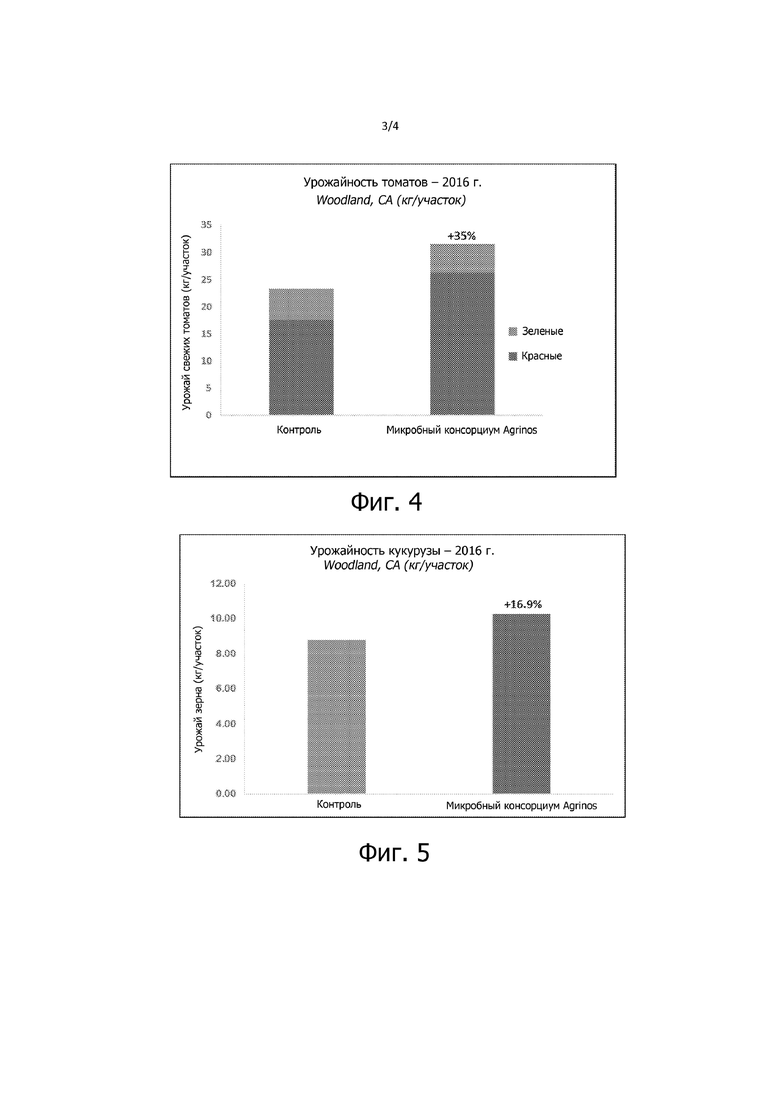
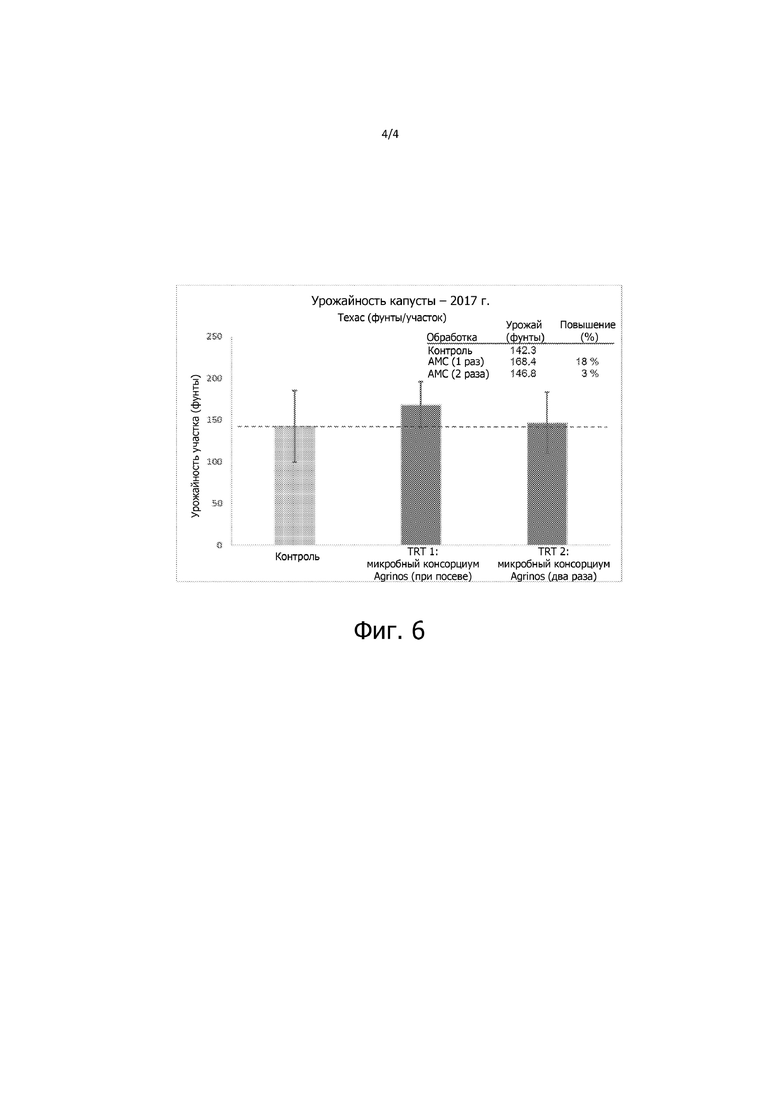
Изобретение относится к композиции для обработки почвы, растений или частей растения и ее применению. Предложена композиция для обработки почвы, растений или частей растения в целях улучшения роста растения, улучшения стрессоустойчивости и повышения урожайности по сравнению с тем, когда почва, растения или части растений не были обработаны. Указанная композиция содержит клетки микроорганизмов, включающих или состоящих из следующих видов: Lactobacillus delbrueckii, Virgibacillus halophilus, Azotobacter vinelandii, Clostridium pasteurianum, Paenibacillus chibensis, Streptomyces griseus, Pseudomonas sp., Pseudomonas putida, Bacillus sp., Bacillus amyloliquefaciens, Oceanobacillus oncorhynchi, Paenibacillus lautus, Bacillus licheniformis, Lactobacillus vini, Paenibacillus cookii, Bacillus subtilis, Lactobacillus buchneri, Bacillus megaterium, Acetobacter pasteurianus, Clostridium beijerinckii, Lactobacillus casei/paracasei и Bacillus flexus. Указанную композицию используют в способе улучшения роста растения, улучшения стрессоустойчивости и повышения урожайности по сравнению с тем, когда почва, растения или части растений не были обработаны композицией. Изобретение обеспечивает улучшение роста растения, улучшение стрессоустойчивости и повышение урожайности. 2 н. и 8 з.п. ф-лы, 6 ил., 13 табл., 7 пр.
1. Композиция для обработки почвы, растений или частей растения в целях улучшения роста растения, улучшения стрессоустойчивости и повышения урожайности по сравнению с тем, когда почва, растения или части растений не были обработаны, содержащая клетки микроорганизмов, включающих или состоящих из следующих видов: Lactobacillus delbrueckii, имеющей нуклеотидную последовательность 16S рДНК, содержащую SEQ ID NO: 24, Virgibacillus halophilus, имеющей нуклеотидную последовательность 16S рДНК, содержащую SEQ ID NO: 23, Azotobacter vinelandii, имеющей нуклеотидную последовательность 16S рДНК, содержащую SEQ ID NO: 22, Clostridium pasteurianum, имеющей нуклеотидную последовательность 16S рДНК, содержащую SEQ ID NO: 21, Paenibacillus chibensis, имеющей нуклеотидную последовательность 16S рДНК, содержащую SEQ ID NO: 19, Streptomyces griseus, имеющей нуклеотидную последовательность 16S рДНК, содержащую SEQ ID NO: 18, Pseudomonas sp., имеющей нуклеотидную последовательность 16S рДНК, содержащую SEQ ID NO: 17, Pseudomonas putida, имеющей нуклеотидную последовательность 16S рДНК, содержащую SEQ ID NO: 16, Bacillus sp., имеющей нуклеотидную последовательность 16S рДНК, содержащую SEQ ID NO: 15, Bacillus amyloliquefaciens, имеющей нуклеотидную последовательность 16S рДНК, содержащую SEQ ID NO: 14, Oceanobacillus oncorhynchi, имеющей нуклеотидную последовательность 16S рДНК, содержащую SEQ ID NO: 13, Paenibacillus lautus, имеющей нуклеотидную последовательность 16S рДНК, содержащую SEQ ID NO: 12, Bacillus licheniformis, имеющей нуклеотидную последовательность 16S рДНК, содержащую SEQ ID NO: 11, Lactobacillus vini, имеющей нуклеотидную последовательность 16S рДНК, содержащую SEQ ID NO: 10, Paenibacillus cookiim, имеющей нуклеотидную последовательность 16S рДНК, содержащую SEQ ID NO: 9, Bacillus subtilis, имеющей нуклеотидную последовательность 16S рДНК, содержащую SEQ ID NO: 8, Lactobacillus buchneri, имеющей нуклеотидную последовательность 16S рДНК, содержащую SEQ ID NO: 7, Bacillus megaterium, имеющей нуклеотидную последовательность 16S рДНК, содержащую SEQ ID NO: 3, Acetobacter pasteurianus, имеющей нуклеотидную последовательность 16S рДНК, содержащую SEQ ID NO: 6, Clostridium beijerinckii, имеющей нуклеотидную последовательность 16S рДНК, содержащую SEQ ID NO: 5, Lactobacillus casei/paracasei, имеющей нуклеотидную последовательность 16S рДНК, содержащую SEQ ID NO: 4, и Bacillus flexus, имеющей нуклеотидную последовательность 16S рДНК, содержащую SEQ ID NO: 20.
2. Композиция по п. 1, дополнительно содержащая клетки одного или нескольких из Desulfosporosinus meridiei, Nitrosopumilus sp., Marinobacter bryozoorum, Leptospirillum ferrodiazotrophum и Lactobacillus acidophilus.
3. Композиция по п. 1 или 2, дополнительно содержащая одно или несколько из хитина, хитозана, глюкозамина, аминокислот и жидких удобрений.
4. Способ улучшения роста растения, улучшения стрессоустойчивости и повышения урожайности по сравнению с тем, когда почва, растения или части растений не были обработаны, включающий контактирование почвы, растений, частей растений или семян с композицией по любому из пп. 1-3.
5. Способ по п. 4, дополнительно включающий контактирование почвы, растений, частей растений или семян с одним или несколькими из хитина, хитозана, глюкозамина и аминокислот.
6. Способ по п. 4 или 5, дополнительно включающий контактирование почвы, растений, частей растений или семян с твердой фракцией, жидкой фракцией или обеими, полученными ферментацией содержащего хитин биологического материала с композицией по любому из пп. 1-3.
7. Способ по любому из пп. 4-6, дополнительно включающий контактирование почвы, растений, частей растений или семян с жидким удобрением.
8. Способ по любому из пп. 4-7, дополнительно включающий контактирование почвы, растений, частей растений или семян с одним или несколькими пестицидами, одним или несколькими фунгицидами, одним или несколькими гербицидами, одним или несколькими инсектицидами, одним или несколькими растительными гормонами, одним или несколькими растительными элиситорами либо комбинациями из двух или нескольких из них.
9. Способ по любому из пп. 4-8, дополнительно включающий активацию микроорганизмов в композиции перед контактированием почвы, растений, частей растений или семян с композицией.
10. Способ по п. 4, включающий контактирование почвы с композицией с получением обработанной почвы и культивирование семян, проростков или растений в обработанной почве.
| WO 2011157747 A2, 22.12.2011 | |||
| Изложница с суживающимся книзу сечением и с вертикально перемещающимся днищем | 1924 |
|
SU2012A1 |
| СПОСОБ ОБРАБОТКИ РАСТЕНИЙ ОГУРЦА ХИТОЗАНОМ ПРОТИВ ГАЛЛОВОЙ НЕМАТОДЫ (MELOIDOGYNE SPP.) | 2007 |
|
RU2353085C1 |
Авторы
Даты
2023-01-16—Публикация
2017-08-30—Подача