Перекрестная ссылка на родственные заявки
[0001] В настоящей заявке испрашивается приоритет предварительной заявки на патент США рег. № 63/229870, поданной 5 августа 2021 г., предварительной заявки на патент США рег. № 63/127648, поданной 18 декабря 2020 г., и предварительной заявки на патент США рег. № 63/093459, поданной 19 октября 2020 г., каждая из которых включена в настоящее описание посредством ссылки.
Заявление о проведении исследований или разработок, спонсируемых Федеральными Органами
[0002] Отсутствует.
Список последовательностей
[0003] Настоящая заявка содержит список последовательностей, который был подан в электронном виде в формате ASCII и в полном объеме включен в настоящее описание посредством ссылки. Указанная копия ASCII, созданная 20 марта 2023 года, имеет название CBI039_30_SL.txt и размер 203259 килобайт.
Область техники
[0004] В целом, настоящее изобретение относится к кластеризованным системам с регулярными чередующимися короткими палиндромными повторами (CRISPR). В частности, настоящее изобретение относится к полинуклеотидам и к направляющим CRISPR для использования в системах CRISPR-Cas12, где полинуклеотиды CRISPR и направляющие Cas12 сконструированы так, что они включают рибонуклеотидные основания и одно или несколько дезоксирибонуклеотидных оснований. Настоящее изобретение дополнительно относится к комплексам направляющий Cas12 /нуклеопротеин, содержащим сконструированный направляющий Cas12 CRISPR и белок CRISPR-Cas12, и к получению модифицированных клеток с использованием таких комплексов направляющий Cas12/нуклеопротеин. Настоящее изобретение также относится к композициям, содержащим полинуклеотиды CRISPR, направляющие Cas12 и комплексы направляющий Cas12/нуклеопротеин, а также к способам их получения и применения. Кроме того, настоящее изобретение относится к получению и терапевтическому применению клеток, модифицированных с использованием комплексов направляющий Cas12/нуклеопротеин согласно изобретению, и, например, к получению клеток, экспрессирующих химерный антигенный рецептор (CAR), для лечения рака.
Предпосылки создания изобретения
[0005] Кластеризованные регулярно чередующиеся короткие палиндромные повторы (CRISPR) и CRISPR-ассоциированные (Cas) белковые системы обнаружены в геномах многих прокариот (бактерий и архебактерий). Эти системы обеспечивают адаптивный иммунитет прокариот против чужеродных патогенов (например, вирусов, бактериофагов). Таким образом, система CRISPR функционирует как разновидность иммунной системы, помогающей защищать прокариоты от чужеродных патогенов. См., например, Barrangou et al. (Science, 2007, 315:1709-1712); Makarova et al. (Nature Reviews Microbiology, 2011, 9:467-477); Garneau et al. (Nature, 2010, 468:67-71); Sapranauskas et al. (Nucleic Acids Research, 2011, 39:9275-9282); Koonin et al. (Curr. Opin. Microbiol., 2017, 37:67-78); Shmakov et al. (Nat. Rev. Microbiol., 2017, 15(3):169-182); Makarova et al. (Nat. Rev. Microbiol., 2020, 18:67-83).
[0005] Действие мммунных систем CRISPR-Cas происходит в три основные стадии: (1) приобретение, (2) экспрессия и (3) интерференция. Приобретение включает расщепление генома инвазивных вирусов и плазмид и интеграцию сегментов (называемых протоспейсерами) геномной ДНК в локус CRISPR организма-хозяина. Сегменты, которые интегрированы в геном хозяина, известны как спейсеры, которые опосредуют защиту от последующей атаки тем же (или достаточно родственным) вирусом или плазмидой. Экспрессия включает транскрипцию локуса CRISPR и последующую ферментативную обработку для получения коротких зрелых РНК CRISPR, каждая из которых содержит одну спейсерную последовательность. Интерференция индуцируется после того, как РНК CRISPR связываются с белками Cas с образованием эффекторных комплексов, которые затем нацеливаются на комплементарные протоспейсеры в чужеродных генетических элементах, что способствует индуцированию разложения нуклеиновых кислот.
[0007] Различные системы CRISPR-Cas в их нативных хозяевах способны к нацеливанию на ДНК (класса 1 типа I; класса 2 типа II и V), нацеливанию на РНК (класса 2 типа VI) и совместному нацеливанию на ДНК и РНК (класса 1 типа III). См., например, Makarova et al. (Nat. Rev. Microbiol., 2015, 13:722-736); Shmakov et al. (Nat. Rev. Microbiol., 2017, 15:169-182); Abudayyeh et al. (Science, 2016, 353: 1-17) Makarova et al. (The CRISPR Journal, 2018, 1:325-336).
[0008] Системы типа V классифицируются на несколько различных подтипов, включая, например, V-A, V-B, V-C, V-D, V-E, V-F, V-G, V-H, V-I, V-J, V-K и V-U. См., например, Makarova et al. (Nat. Rev. Microbiol., 2020, 18:67-83) и Pausch et al. (Science, 2020, 369(6501):333-337). Подтип V-A кодирует белок Cas12a (ранее известный как Cpf1). Cas12a имеет RuvC-подобный домен нуклеазы, который гомологичен соответствующему домену Cas9, но не имеет домена нуклеазы HNH, который присутствует в белках Cas9.
[0009] Системы типа V были идентифицированы у нескольких бактерий, включая бактерию Parcubacteria GWC2011_GWC2_44_17 (PbCpf1), бактерию Lachnospiraceae MC2017 (Lb3 Cpf1), Butyrivibrio proteoclasticus (BpCpf1), бактерию Peregrinibacteria GW2011_GWA_33_10 (PeCpf1), Acidaminococcus sp. BV3L6 (AsCpf1), Porphyromonas macacae (PmCpf1), бактерию Lachnospiraceae ND2006 (LbCpf1), Porphyromonas crevioricanis (PcCpf1), Prevotella disiens (PdCpf1), Moraxella bovoculi 237 (MbCpf1), Smithella sp. SC_K08D17 (SsCpf1), Leptospira inadai (LiCpf1), бактерию Lachnospiraceae MA2020 (Lb2Cpf1), Franciscella novicida U112 (FnCpf1), Candidatus methanoplasma termitum (CMtCpf1) и Eubacterium eligens (EeCpf1).
[0010] Системы CRISPR-Cas представляют собой мощные инструменты для сайт-направленного редактирования генома посредством удаления, инсерции, мутации или замены специфических последовательностей нуклеиновых кислот. Изменение может быть специфичным для гена или для участка локализации. Редактирование генома может быть осуществлено посредством сайт-направленных нуклеаз, таких как Cas-белки и родственные им полинуклеотиды, для разрезания нуклеиновой кислоты-мишени, с последующим созданием сайта для модификации. В некоторых случаях, расщепление может приводить к двухцепочечному разрыву (DSB) в последовательности ДНК-мишени. DSB могут быть подвергнуты репарации, например, посредством негомологичного соединения концов (NHEJ), опосредованного микрогомологией соединения концов (MMEJ) или репарации на основе гомологии (HDR). HDR зависит от наличия матрицы для репарации. В некоторых примерах такого редактирования генома, донорный полинуклеотид или его часть могут быть встроены в разрыв.
Описание сущности изобретения
[0011] Настоящее изобретение основано на открытии новых полинуклеотидов и направляющих последовательностей для использования в системах CRISPR-Cas типа V, полинуклеотидов и направляющих последовательностей, содержащих рибонуклеотидные основания и одно или несколько дезоксирибонуклеотидных оснований. Раскрытые здесь направляющие последовательности, при образовании комплекса с белком CRISPR-Cas типа V, таким как Cas12a, способны к надежному редактированию генома-мишени и к уменьшенному уровню редактирования генома, не являющегося мишенью.
[0012] Этот процесс редактирования генома является особенно подходящим для получения генетически модифицированных клеток, используемых в терапевтических целях. Так, например, с помощью такого процесса редактирования генома, иммунные клетки (такие как Т-клетки) могут быть генетически модифицированы для экспрессии CAR. Такие CAR-экспрессирующие клетки могут быть использованы, например, в адоптивной иммунотерапии, где CAR-экспрессирующие иммунные клетки, такие как Т-клетки (CAR-T-клетки), могут вводиться пациентам для нацеливания на клетки, экспрессирующие антиген-мишень, распознаваемый CAR (например, чужеродный антиген или ассоциированный с раком антиген).
[0013] Неограничивающими вариантами осуществления изобретения являются нижеследующие варианты.
[0014] [1] Направляющая молекула CRISPR, содержащая: нацеливающую область, способную связываться с последовательностью нуклеиновых кислот-мишеней; и активирующую область, способную образовывать нуклеопротеиновый комплекс с белком Cas12, где указанная направляющая молекула CRISPR содержит рибонуклеотидные основания и по меньшей мере одно дезоксирибонуклеотидное основание.
[0015] [2] Направляющая молекула CRISPR по [1], где направляющая молекула CRISPR содержит по меньшей мере одно дезоксирибонуклеотидное основание в активирующей области, нацеливающей области или в обеих областях.
[0015] [3] Направляющая молекула CRISPR по [1], где направляющая молекула CRISPR дополнительно содержит один или более аналогов оснований, выбранных из группы, состоящей из инозина, дезоксиинозина, дезоксиурацила, ксантозина, спейсера C3, 5-метил-dC, 5-гидроксибутинил-2'-дезоксиуридина, 5-нитроиндола, 5-метилизодезоксицитозина, изодезоксигуанозина, дезоксиуридина и изодезоксицитидина.
[0017] [4] Направляющая молекула CRISPR по [1], где направляющая молекула CRISPR дополнительно содержит один или несколько неосновных сайтов.
[0018] [5] Направляющая молекула CRISPR по [1], где направляющая молекула CRISPR выбрана из: направляющей молекулы CRISPR, содержащей последовательность РНК UAAUUUCUACUUCUUGUAGAUGAGUCUCUCAGCUGGUACAC, где по меньшей мере одно из оснований в последовательности заменено соответствующим дезоксирибонуклеотидным основанием, и, необязательно, по меньшей мере одно из оснований в последовательности заменено аналогом основания или неосновным сайтом; и направляющей молекулы CRISPR, содержащей последовательность РНК UAAUUUCUACUUCUUGUAGAUAGUAGUGGGGGUGGUGAUUCAGUGU, где по меньшей мере одно из оснований в последовательности заменено на соответствующее дезоксирибонуклеотидное основание, и необязательно, по меньшей мере одно из оснований в последовательности заменено аналогом основания или неосновным сайтом.
[0019] [6] Направляющая молекула CRISPR по [5], где количество дезоксирибонуклеотидных оснований в направляющей молекуле CRISPR в процентах от общего размера направляющей молекулы CRISPR составляет 50% или менее.
[0020] [7] Направляющая молекула CRISPR по [6], где количество дезоксирибонуклеотидных оснований в направляющей молекуле CRISPR в процентах от общего размера направляющей молекулы составляет 25% или менее.
[0021] [8] Направляющая молекула CRISPR по [5], где количество дезоксирибонуклеотидных оснований в нацеливающей области в процентах от общего размера нацеливающей области составляет 25% или менее.
[0022] [9] Направляющая молекула CRISPR по [8], где количество дезоксирибонуклеотидных оснований в нацеливающей области в процентах от общего размера нацеливающей области составляет 5% или менее.
[0023] [10] Направляющая молекула CRISPR по [5], где количество дезоксирибонуклеотидных оснований в активирующей области в процентах от общего размера активирующей области составляет 50% или менее.
[0024] [11] Направляющая молекула CRISPR по [10], где количество дезоксирибонуклеотидных оснований в активирующей области в процентах от общего размера активирующей области составляет 25% или менее.
[0025] [12] Направляющая молекула CRISPR по [5], где направляющая молекула CRISPR обладает пониженной нецелевой активностью по сравнению с направляющей молекулой CRISPR, содержащей только РНК, которая связывается с последовательностью нуклеиновой кислоты-мишени, и которая способна образовывать нуклеопротеиновый комплекс с белком Cas12.
[0025] [13] Направляющая молекула CRISPR по [5], где одно или несколько положений 1, 3, 7, 10, 12, 14, 15, 16, 17, 18, 19, 21, 28, 29, 30, 31, 32, 34, 35, 36, 37, 38, 39 и 40 в последовательности содержат дезоксирибонуклеотидное основание.
[0027] [14] Направляющая молекула CRISPR по [13], где пятнадцать или менее положений 1, 3, 7, 10, 12, 14, 15, 16, 17, 18, 19, 21, 28, 29, 30, 31, 32, 34, 35, 36, 37, 38, 39 и 40 в последовательности содержат дезоксирибонуклеотидные основания.
[0028] [15] Направляющая молекула CRISPR по [14], где двенадцать или менее положений 1, 3, 7, 10, 12, 14, 15, 16, 17, 18, 19, 21, 28, 29, 30, 31, 32, 34, 35, 36, 37, 38, 39 и 40 в последовательности содержат дезоксирибонуклеотидные основания.
[0029] [16] Направляющая молекула CRISPR по [2], где направляющая молекула CRISPR содержит активирующую область, содержащую последовательность РНК UAAUUUCUACUUCUUGUAGAU, где по меньшей мере одно из оснований в последовательности заменено соответствующим дезоксирибонуклеотидным основанием, и необязательно, по меньшей мере одно из оснований в последовательности заменено аналогом основания или неосновным сайтом.
[0030] [17] Направляющая молекула CRISPR по [16], где одно или несколько положений 1, 3, 7, 10, 12, 14, 15, 16, 17, 18, и 19 в последовательности содержат дезоксирибонуклеотидное основание.
[0031] [18] Направляющая молекула CRISPR по [17], где десять или менее положений 1, 3, 7, 10, 12, 14, 15, 16, 17, 18, и 19 в последовательности содержат дезоксирибонуклеотидные основания.
[0032] [19] Направляющая молекула CRISPR по [18], где восемь или менее положений 1, 3, 7, 10, 12, 14, 15, 16, 17, 18, и 19 в последовательности содержат дезоксирибонуклеотидные основания.
[0033] [20] Направляющая молекула CRISPR по [2], где направляющая молекула CRISPR содержит нацеливающую область, содержащую последовательность РНК GAGUCUCUCAGCUGGUACAC, где по меньшей мере одно из оснований в последовательности заменено соответствующим дезоксирибонуклеотидным основанием, и необязательно, по меньшей мере одно из оснований в последовательности заменено на аналог основания или неосновный сайт.
[0034] [21] Направляющая молекула CRISPR по [20], где одно или несколько положений 1, 8, 9, 10, 11, 12, 14, 15, 16, 17, 18, 19, и 20 в последовательности содержат дезоксирибонуклеотидное основание.
[0035] [22] Направляющая молекула CRISPR по [21], где пять или менее положений 1, 8, 9, 10, 11, 12, 14, 15, 16, 17, 18, 19 и 20 в последовательности содержат дезоксирибонуклеотидные основания.
[0035] [23] Направляющая молекула CRISPR по [22], где три или менее положений 1, 8, 9, 10, 11, 12, 14, 15, 16, 17, 18, 19 и 20 в последовательности содержат дезоксирибонуклеотидные основания.
[0037] [24] Направляющая молекула CRISPR по [2], где направляющая молекула CRISPR содержит нацеливающую область, содержащую последовательность РНК AGUGGGGGUGAAUUCAGUGU, где по меньшей мере одно из оснований в последовательности заменено соответствующим дезоксирибонуклеотидным основанием, и необязательно, по меньшей мере одно из оснований в последовательности заменено на аналог основания или неосновный сайт.
[0038] [25] Направляющая молекула CRISPR по [24], где одно или несколько положений 1, 8, 9, 10, 11, 12, 14, 15, 16, 17, 18, 19 и 20 в последовательности содержат дезоксирибонуклеотидное основание.
[0039] [26] Направляющая молекула CRISPR по [25], где пять или менее положений 1, 8, 9, 10, 11, 12, 14, 15, 16, 17, 18, 19 и 20 в последовательности содержат дезоксирибонуклеотидные основания.
[0040] [27] Направляющая молекула CRISPR по [26], где три или менее положений 1, 8, 9, 10, 11, 12, 14, 15, 16, 17, 18, 19 и 20 в последовательности содержат дезоксирибонуклеотидные основания.
[0041] [28] Направляющая молекула CRISPR по [5], где направляющая молекула CRISPR содержит последовательность TAAUUUCUACUCUTGUAGAUGAGUCUCUCAGCUGGUACAC, где положения 2, 4, 5, 6, 8, 9, 11, 13, 16, 17, 18, 20, 22, 23, 24, 25, 26, 27, 28, 29, 30, 32, 33, 34, 35, 36, 37, 38 и 39 в последовательности содержат рибонуклеотидные основания, и где положения 1, 3, 7, 10, 12, 14, 15, 19, 21, 31 и 40 в последовательности содержат дезоксирибонуклеотидные основания.
[0042] [29] Направляющая молекула CRISPR по [5], где направляющая молекула CRISPR содержит последовательность TAAUUUCUACUCUTGUAGAUAGUGGGGGUGAUUCAGUGT, где положения 2, 4, 5, 6, 8, 9, 11, 13, 16, 17, 18, 20, 22, 23, 24, 25, 26, 27, 28, 29, 30, 32, 33, 34, 35, 36, 37, 38 и 39 в последовательности содержат рибонуклеотидные основания, и где положения 1, 3, 7, 10, 12, 14, 15, 19, 21, 31 и 40 в последовательности содержат дезоксирибонуклеотидные основания.
[0043] [30] Направляющая молекула CRISPR по [2], где активирующая область имеет длину 20 оснований, и где одно или более положений 1, 3, 7, 10, 12, 14, 15, 16, 17, 18 и 19 в последовательности активирующей области из 20 нуклеотидов включают дезоксирибонуклеотидное основание.
[0044] [31] Направляющая молекула CRISPR по [30], где десять или менее положений 1, 3, 7, 10, 12, 14, 15, 16, 17, 18 и 19 в последовательности содержат дезоксирибонуклеотидные основания.
[0045] [32] Направляющая молекула CRISPR по [31], где восемь или менее положений 1, 3, 7, 10, 12, 14, 15, 16, 17, 18 и 19 в последовательности содержат дезоксирибонуклеотидные основания.
[0045] [33] Направляющая молекула CRISPR по [2], где нацеливающая область имеет длину 20 оснований и где одно или более положений 1, 8, 9, 10, 11, 12, 14, 15, 16, 17, 18, 19 и 20 в последовательности нацеливающей области из 20 нуклеотидов содержат дезоксирибонуклеотидное основание.
[0047] [34] Направляющая молекула CRISPR по [33], где пять или менее положений 1, 8, 9, 10, 11, 12, 14, 15, 16, 17, 18, 19 и 20 в последовательности содержат дезоксирибонуклеотидные основания.
[0048] [35] Направляющая молекула CRISPR по [34], где три или менее положений 1, 8, 9, 10, 11, 12, 14, 15, 16, 17, 18, 19 и 20 в последовательности содержат дезоксирибонуклеотидные основания.
[0049] [36] Композиция нуклеиновой кислоты/белка CRISPR, включающая: направляющую молекулу CRISPR по любому из [1]-[35]; и белок Cas12.
[0050] [37] Композиция нуклеиновой кислоты/белка CRISPR по [36], где направляющая молекула CRISPR находится в комплексе с белком Cas12.
[0051] [38] Композиция нуклеиновой кислоты/белка CRISPR по [36], где белок Cas12 представляет собой белок Cas12a.
[0052] [39] Композиция нуклеиновой кислоты/белка CRISPR по любому из [36]-[38], где белок Cas12 содержит у С-конца последовательность, содержащую линкер и NLS, которая по меньшей мере на 80% идентична аминокислотной последовательности, выбранной из группы, состоящей из SEQ ID NO: 479-490.
[0053] [40] Композиция нуклеиновой кислоты/белка CRISPR по [39], где последовательность, содержащая линкер и NLS, включает аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 483, 485, 487 и 489.
[0054] [41] Клетка, содержащая направляющую молекулу CRISPR по любому из [1]-[35].
[0055] [42] Клетка по [41], дополнительно содержащая белок Cas12.
[0055] [43] Клетка по [42], где белок Cas12 представляет собой белок Cas12a.
[0057] [44] Клетка по [42] или [43], где белок Cas12 содержит у С-конца последовательность, которая содержит линкер и NLS, и которая по меньшей мере на 80% идентична аминокислотной последовательности, выбранной из группы, состоящей из SEQ ID NO: 479-490.
[0058] [45] Клетка по [44], где последовательность, содержащая линкер и NLS, включает аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 483, 485, 487 и 489.
[0059] [46] Клетка по любому из [42]-[45], где направляющая молекула CRISPR находится в комплексе с белком Cas12.
[0060] [47] Клетка по любому из [41]-[46], где клетка представляет собой прокариотическую клетку или эукариотическую клетку.
[0061] [48] Клетка по [47], где клетка представляет собой эукариотическую клетку, выбранную из группы, состоящей из одноклеточного эукариотического организма, клетки эукариотического организма, клетки простейших, клетки растения, клетки водорослей, клетки грибов, клетки животного, клетки беспозвоночного животного, клетки позвоночного животного, клетки млекопитающего, стволовой клетки и клетки-предшественника.
[0062] [49] Клетка по [48], где клетка представляет собой лимфоцит, Т-клетку, содержащую химерный антигенный рецептор (CAR), клетку, содержащую Т-клеточнЫЙ рецептор (TCR), CAR-T-клетку, сконструированную на основе TCR, инфильтрирующий опухоль лимфоцит (TIL), CAR TIL, дендритную клетку (DC), CAR-DC, макрофаг, CAR-макрофаг (CAR-M), природную клетку-киллер (NK) или CAR-NK-клетку.
[0063] [50] Клетка по [49], где клетка представляет собой CAR-T-клетку.
[0064] [51] Клетка по любому из [41]-[50], дополнительно содержащая донорный полинуклеотид.
[0065] [52] Способ расщепления последовательности нуклеиновой кислоты-мишени, где указанный способ включает: контактирование первой последовательности нуклеиновой кислоты-мишени с нуклеопротеиновым комплексом, включающим каталитически активный белок Cas12 и первую направляющую молекулу CRISPR, где первая направляющая молекула CRISPR содержит направляющую молекулу CRISPR по любому из [1]-[35]; где нацеливающая область первой направляющей молекулы CRISPR способна к гибридизации с первой последовательностью нуклеиновой кислоты-мишени, а нуклеопротеиновый комплекс способен расщеплять первую последовательность нуклеиновой кислоты-мишени.
[0065] [53] Способ по [52], дополнительно включающий получение донорного полинуклеотида.
[0067] [54] Способ по [53], где последовательность нуклеиновой кислоты-мишени расщепляется с образованием сайта расщепления, и где способ дополнительно включает модификацию последовательности нуклеиновой кислоты-мишени.
[0068] [55] Способ по [54], где модификация включает введение по меньшей мере части донорного полинуклеотида в сайт расщепления.
[0069] [56] Способ по [54], где модификация включает удаление одного или более нуклеотидов в сайте расщепления.
[0070] [57] Способ по [55], где последовательность нуклеиновых кислот-мишеней находится в клетке.
[0071] [58] Способ по [57], где клетка содержит эукариотическую клетку.
[0072] [59] Способ по [58], где донорный полинуклеотид содержит вектор для экспрессии CAR.
[0073] [60] Способ по [59], дополнительно включающий введение вектора для экспрессии CAR в клетку с использованием вирусного вектора.
[0074] [61] Способ по [60], где указанное введение включает трансдукцию.
[0075] [62] Способ по любому из [57]-[61], где полученная клетка содержит лимфоцит, CAR-T-клетку, TCR-клетку, CAR-T-клетку, сконструированную на основе TCR, TIL, CAR-TIL, дендритную клетку, CAR-DC, макрофаг, CAR-M, NK-клетку или CAR-NK-клетку.
[0075] [63] Способ по любому из [52]-[62], где первая последовательность нуклеиновой кислоты-мишени находится внутри гена-мишени, кодирующего белок, выбранный из группы, состоящей из TRAC; TRBV; бета-2-микроглобулина (B2M); PD1; PD-L1; CTLA-4; LAG-3; TIGIT; TIM3; HLA-E; HLA-A; HLA-B; HLA-C; HLA-DRA; ADAM17; BTLA; CD160; SIGLEC10; 2B4; LAIR1; CD52; CD96; VSIR; VISTA; KIR2DL1; KIR2DL2; KIR2DL3; CEACAM1; CBLB; CISH; IL-1R8; AHR; рецептора аденозина 2A; GMCSF; VISTA; CII2A; и NKG2A.
[0077] [64] Способ по любому из [59]-[61], где вектор для экспрессии CAR кодирует CAR, содержащий внеклеточный лиганд-связывающий домен.
[0078] [65] Способ по [64], где вектор для экспрессии CAR дополнительно кодирует шарнирную область, трансмембранную область и одну или несколько внутриклеточных сигнальных областей.
[0079] [66] Способ по [64] или [65], где внеклеточный лиганд-связывающий домен содержит вариабельный одноцепочечный фрагмент иммуноглобулина (scFv).
[0080] [67] Способ по [66], где scFv способен связываться с клеточной мишенью, выбранной из группы, состоящей из CD37, CD38, CD47, CD73, CD4, CS1, PD-L1, NGFR, ENPP3, PSCA, CD79B, TACI, VEGFR2, B7-H3, B7-H6, антигена созревания В-клеток (BCMA), CD123, CD138, CD171/L1CAM, CD19, CD20, CD22, CD30, CD33, CD70, CD371, СЕА, клаудина 18.1, клаудина 18.2, CSPG4, EFGRvIII, EpCAM, EphA2, рецептора эпидермального фактора роста, ErbB, ErbB2 (HER2), FAP, FRa, GD2, GD3, глипикана 3, IL-11Ra, IL-13Ra2, рецептора альфа IL13, антигена Льюиса Y/LeY, мезотелина, MUC1, MUC16, лигандов NKG2D, PD1, PSMA, ROR-1, SLAMF7, TAG72, ULBP и белков MICA/B, VEGF2 и WT1.
[0081] [68] Способ по [67], где scFv способен связываться с клеточной мишенью, выбранной из группы, состоящей из BCMA, CD19, CD20, CD22, CD47, CD371, ROR-1, EphA2, MUC16, глипикана 3, PSCA и клаудина 18.2.
[0082] [69] Способ по [68], где scFv способен связываться с BCMA.
[0083] [70] Способ по [68], где scFv способен связываться с CD371.
[0084] [71] Способ по любому из [57]-[62] и [64]-[70], где указанный способ дополнительно включает контактирование второй последовательности нуклеиновой кислоты-мишени в клетке с нуклеопротеиновым комплексом, включающим каталитически активный белок Cas12 и вторую направляющую молекулу CRISPR, где вторая направляющая молекула CRISPR содержит направляющую молекулу CRISPR по любому из [1]-[35], которая способна связываться с другой последовательностью нуклеиновой кислоты-мишени, отличающейся от первой направляющей молекулы CRISPR, где нацеливающая область второй направляющей молекулы CRISPR способна гибридизоваться со второй последовательностью нуклеиновой кислоты-мишени, и нуклеопротеиновый комплекс способен расщеплять вторую последовательность нуклеиновой кислоты-мишени.
[0085] [72] Способ по [71], где указанные первая и вторая последовательности нуклеиновых кислот-мишеней находятся независимо внутри гена-мишени, кодирующего белок, выбранный из группы, состоящей из TRAC; белка TRBV; бета-2-микроглобулина (B2M); PD1; PD-L1; CTLA-4; LAG-3; TIGIT; TIM3; HLA-E; HLA-A; HLA-B; HLA-C; HLA-DRA; ADAM17; BTLA; CD160; SIGLEC10; 2B4; LAIR1; CD52; CD96; VSIR; VISTA; KIR2DL1; KIR2DL2; KIR2DL3; CEACAM1; CBLB; CISH; IL-1R8; AHR; рецептора аденозина 2A; GMCSF; VISTA; CII2A; и NKG2A.
[0085] [73] Способ по [71] или [72], где донорный полинуклеотид содержит вектор для экспрессии CAR, где CAR содержит внеклеточный лигандсвязывающий домен, и где внеклеточный лигандсвязывающий домен содержит scFv.
[0087] [74] Способ по [73], где scFv способен связываться с BCMA.
[0088] [75] Способ по [73], где scFv способен связываться с CD371.
[0089] [78] Способ по [72], где указанная первая последовательность нуклеиновой кислоты-мишени находится внутри гена, кодирующего белок TRAC, и где указанная вторая последовательность нуклеиновой кислоты-мишени находится внутри гена, кодирующего белок PD1.
[0090] [77] Способ по [72], где указанная первая последовательность нуклеиновой кислоты-мишени находится внутри гена, кодирующего белок TRAC, и где указанная вторая последовательность нуклеиновой кислоты-мишени находится внутри гена, кодирующего белок B2M.
[0091] [78] Способ по [77], дополнительно включающий введение в клетку второго донорного полинуклеотида, содержащего конструкцию для слияния B2M-HLA-E, и где по меньшей мере часть второго донорного полинуклеотида, содержащего конструкцию для слияния B2M-HLA-E, вводят в сайт расщепления второй последовательности нуклеиновой кислоты-мишени, где конструкция для слияния B2M-HLA-E кодирует слитый белок, содержащий, от N- до С-конца, сигнал секреции B2M, сигнальную последовательность пептида HLA-G, первую линкерную последовательность, последовательность B2M, вторую линкерную последовательность и последовательность HLA-E.
[0092] [79] Способ по [69] или [74], где анти-BCMA scFv содержит вариабельную область тяжелой цепи (VH), содержащую аминокислотную последовательность SEQ ID NO:474; и вариабельную область легкой цепи (VL), содержащую аминокислотную последовательность SEQ ID NO: 475.
[0093] [80] Способ по [79], где scFv дополнительно содержит линкер между VH и VL.
[0094] [81] Способ по [80], где линкер содержит аминокислотную последовательность SEQ ID NO:476.
[0095] [82] Способ по [81], где указанный scFv содержит аминокислотную последовательность SEQ ID NO:477.
[0095] [83] Способ по [64] или [73], где CAR содержит: scFv, включающий VH и VL; трансмембранный домен; костимулирующий домен; и активирующий домен.
[0097] [84] Способ по [83], где трансмембранный домен представляет собой трансмембранный домен, происходящий от α-цепи Т-клеточного рецептора, β-цепи Т-клеточного рецептора, цепи CD3ζ, CD28, CD3ε, CD45, CD4, CD5, CD8, CD9, CD16, CD22, CD33, CD37, CD64, CD80, CD86, CD134, CD137, ICOS, CD154 или GITR.
[0098] [85] Способ по [84], где трансмембранный домен содержит трансмембранный домен, происходящий от CD8.
[0099] [86] Способ по [83], где костимулирующий домен представляет собой костимулирующий домен, происходящий от CD28, 4-1BB, GITR, ICOS-1, CD27, OX-40 или DAP10.
[00100] [87] Способ по [86], где костимулирующий домен содержит костимулирующий домен 4-1BB.
[00101] [88] Способ по [83], где активирующий домен содержит активирующий домен CD3ζ.
[00102] [89] Способ по [83], где трансмембранный домен содержит трансмембранный домен, происходящий от CD8, костимулирующий домен содержит костимулирующий домен 4-1BB, а активирующий домен содержит активирующий домен CD3ζ.
[00103] [90] Способ по [83], где VH содержит аминокислотную последовательность SEQ ID NO:474, а VL содержит аминокислотную последовательность SEQ ID NO:475.
[00104] [91] Способ по любому из [59]-[62], [64]-[70], [73]-[75] и [78]-[90], где полинуклеотидная последовательность, кодирующая CAR в указанном векторе для экспрессии CAR, имеет лидерную последовательность на 5'-конце.
[00105] [92] Способ по [91], где лидерная последовательность содержит последовательность нуклеиновой кислоты SEQ ID NO:478.
[00105] [93] Способ по [91], где вектор для экспрессии CAR содержит промотор.
[00107] [94] Способ по [93], где промотор содержит промотор MND.
[00108] [95] Способ по любому из [52]-[94], где белок Cas12 содержит у С-конца последовательность, которая включает линкер и NLS, и которая по меньшей мере на 80% идентична аминокислотной последовательности, выбранной из группы, состоящей из SEQ ID NO: 479-490.
[00109] [96] Способ по [95], где последовательность, содержащая линкер и NLS, включает аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 483, 485, 487 и 489.
[00110] [97] Клетка, полученная способом по любому из [57]-[96].
[00111] [98] CAR-T-клетка, полученная способом по любому из [59]-[96].
[00112] [99] CAR-T-клетка по [98], где указанная CAR-T-клетка является аллогенной CAR-T-клеткой.
[00113] [100] CAR-T-клетка по [98], где указанная CAR-T-клетка является аутологичной CAR-T-клеткой.
[00114] [101] Способ получения CAR-T-клетки, включающий осуществление способа по любому из [59]-[96] с использованием Т-лимфоцита в качестве клетки.
[00115] [102] Способ адоптивной клеточной терапии, включающий введение индивидууму, нуждающемуся в этом, клетки, полученной способом по любому из [57]-[96].
[00115] [103] Способ адоптивной клеточной терапии, включающий введение индивидууму, нуждающемуся в этом, CAR-T-клетки, полученной способом по любому из [59]-[96].
[00117] [104] Способ уничтожения BCMA-позитивных раковых клеток, где указанный способ включает контактирование BCMA-позитивных раковых клеток с CAR-T-клетками, полученными способом по любому из [69], [74] и [90].
[00118] [105] Способ по [104], где BCMA-позитивные раковые клетки содержат раковые клетки множественной миеломы.
[00119] [106] Способ по [105], где раковые клетки множественной миеломы содержат клетки человека.
[00120] [107] Способ по [104], где контактирование является внутриопухолевым.
[00121] [108] Способ получения CAR-экспрессирующей клетки, где указанный способ включает: контактирование первой последовательности нуклеиновой кислоты-мишени в клетке с нуклеопротеиновым комплексом, включающим каталитически активный белок Cas12 и первую направляющую молекулу CRISPR, где первая направляющая молекула CRISPR содержит направляющую молекулу CRISPR по любому из [1]-[35], где нацеливающая область первой направляющей молекулы CRISPR способна к гибридизации с первой последовательностью нуклеиновой кислоты-мишени, а нуклеопротеиновый комплекс способен расщеплять первую последовательность нуклеиновой кислоты-мишени; контактирование второй последовательности нуклеиновой кислоты-мишени в клетке с нуклеопротеиновым комплексом, включающим каталитически активный белок Cas12 и вторую направляющую молекулу CRISPR, где вторая направляющая молекула CRISPR содержит направляющую молекулу CRISPR по любому из [1]-[35], которая способна связываться с другой последовательностью нуклеиновой кислоты-мишени, отличающейся от первой направляющей молекулы CRISPR, где нацеливающая область второй направляющей молекулы CRISPR способна к гибридизации со второй последовательностью нуклеиновой кислоты-мишени, а нуклеопротеиновый комплекс способен расщеплять вторую последовательность нуклеиновой кислоты-мишени; и введение в указанную клетку донорного полинуклеотида, содержащего вектор для экспрессии CAR, где по меньшей мере часть донорного полинуклеотида, содержащего указанный вектор для экспрессии CAR, может быть встроена в сайт расщепления в указанной первой последовательности нуклеиновой кислоты-мишени, и где CAR содержит внеклеточный лиганд-связывающий домен.
[00122] [109] Способ по [108], где донорный полинуклеотид, содержащий вектор для экспрессии CAR, вводят в клетку с использованием вирусного вектора.
[00123] [110] Способ по [108], где вектор для экспрессии CAR дополнительно кодирует шарнирную область, трансмембранную область и одну или несколько внутриклеточных сигнальных областей.
[00124] [111] Способ по любому из [108]-[110], где указанная первая последовательность нуклеиновой кислоты-мишени находится внутри гена, кодирующего белок TRAC, и где указанная вторая последовательность нуклеиновой кислоты-мишени находится внутри гена, кодирующего белок PD1.
[00125] [112] Способ по любому из [108]-[110], где указанная первая последовательность нуклеиновой кислоты-мишени находится внутри гена, кодирующего белок TRAC, и где указанная вторая последовательность нуклеиновой кислоты-мишени находится внутри гена, кодирующего белок B2M.
[00125] [113] Способ по любому из [108]-[112], где внеклеточный лиганд-связывающий домен содержит вариабельный одноцепочечный фрагмент иммуноглобулина (scFv).
[00127] [114] Способ по [113], где scFv способен связываться с BCMA.
[00128] [115] Способ по [113], где scFv способен связываться с CD371.
[00129] [116] Способ по [114], где анти-BCMA scFv содержит вариабельную область тяжелой цепи (VH), содержащую аминокислотную последовательность SEQ ID NO:474, и вариабельную область легкой цепи (VL), содержащую аминокислотную последовательность SEQ ID NO:475.
[00130] [117] Способ по [116], где scFv дополнительно содержит линкер между VH и VL.
[00131] [118] Способ по [117], где линкер содержит аминокислотную последовательность SEQ ID NO:476.
[00132] [119] Способ по [114], где указанный scFv содержит аминокислотную последовательность SEQ ID NO:477.
[00133] [120] Способ по любому из [108]-[119], дополнительно включающий введение в клетку второго донорного полинуклеотида, содержащего конструкцию для слияния B2M-HLA-E, где по меньшей мере часть второго донорного полинуклеотида, содержащего конструкцию для слияния B2M-HLA-E, вводят в сайт расщепления второй последовательности нуклеиновой кислоты-мишени, где конструкция для слияния B2M-HLA-E кодирует слитый белок, содержащий, от N- до С-конца, сигнал секреции B2M, сигнальную последовательность пептида HLA-G, первую линкерную последовательность, последовательность B2M, вторую линкерную последовательность и последовательность HLA-E.
[00134] [121] Способ по любому из [108]-[120], где CAR содержит: scFv, содержащий VH и VL; трансмембранный домен; костимулирующий домен; и активирующий домен.
[00135] [122] Способ по [121], где трансмембранный домен содержит трансмембранный домен, происходящий от α-цепи Т-клеточного рецептора, β-цепи Т-клеточного рецептора, цепи CD3ζ, CD28, CD3ε, CD45, CD4, CD5, CD8, CD9, CD16, CD22, CD33, CD37, CD64, CD80, CD86, CD134, CD137, ICOS, CD154 или GITR.
[00135] [123] Способ по [122], где трансмембранный домен содержит трансмембранный домен, происходящий от CD8.
[00137] [124] Способ по [121], где костимулирующий домен содержит костимулирующий домен, происходящий от CD28, 4-1BB, GITR, ICOS-1, CD27, OX-40 или DAP10.
[00138] [125] Способ по [124], где костимулирующий домен содержит костимулирующий домен 4-1BB.
[00139] [126] Способ по [121], где активирующий домен содержит активирующий домен CD3ζ.
[00140] [127] Способ по [121], где трансмембранный домен содержит трансмембранный домен, происходящий от CD8, костимулирующий домен содержит костимулирующий домен 4-1BB, а активирующий домен содержит активирующий домен CD3ζ.
[00141] [128] Способ по любому из [108]-[127], где CAR-экспрессирующая клетка представляет собой CAR-T-клетку.
[00142] [129] Способ по [128], где CAR-T-клетка представляет собой аллогенную CAR-T-клетку.
[00143] [130] Способ по [128], где CAR-T-клетка представляет собой аутологичную CAR-T-клетку.
[00144] [131] Способ по любому из [108]-[130], где белок Cas12, образующий комплекс с первой направляющей молекулой CRISPR и/или белок Cas12, образующий комплекс со второй направляющей молекулой CRISPR, содержит у С-конца последовательность, которая включает линкер и NLS, и которая по меньшей мере на 80% идентична аминокислотной последовательности, выбранной из группы, состоящей из SEQ ID NO: 479-490.
[00145] [132] Способ по [131], где белок Cas12, образующий комплекс с первой направляющей молекулой CRISPR и/или белок Cas12, образующий комплекс со второй направляющей молекулой CRISPR, содержит аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 483, 485, 487 и 489.
[00145] [133] Способ по [78], где второй донорный полинуклеотид дополнительно содержит последовательность P2A на N-конце конструкции для слияния B2M-HLA-E.
[00147] [134] Способ по [120], где второй донорный полинуклеотид дополнительно содержит последовательность P2A на N-конце последовательности конструкции для слияния B2M-HLA-E.
[00148] В некоторых своих вариантах, настоящее изобретение относится к направляющей молекуле CRISPR, содержащей нацеливающую область, способную связываться с последовательностью нуклеиновой кислоты-мишени, и активирующую область, содержащую последовательность РНК UAAUUUCUACUUCUUGUAGAU, включающую по меньшей мере один дезоксирибонуклеотид вместо рибонуклеотида, где активирующая область способна образовывать нуклеопротеиновый комплекс с белком Cas12. В некоторых вариантах осуществления изобретения, одно или более (например, десять или менее) положений 1, 3, 7, 10, 12, 14, 15, 16, 17, 18 и 19 в активирующей области содержат дезоксирибонуклеотидное основание. В некоторых вариантах осуществления изобретения, молекула содержит одну или более химических модификаций, выбранных из группы, состоящей из модификаций оснований, включая инозин, дезоксиинозин, дезоксиурацил, ксантозин, спейсер C3, 5-метил-dC, 5-гидроксибутинил-2'-дезоксиуридин, 5-нитроиндол, 5-метилизодезоксицитозин, изодезоксигуанозин, дезоксиуридин, изодезоксицитидин и неосновный сайт, и модификации остова, включая фосфортиоатную модификацию.
[00149] В некоторых вариантах осуществления изобретения, нацеливающая область направляющей последовательности CRISPR нацелена на ген В2М и содержит последовательность РНК AGUGGGGGUGAAUUCAGUGU, где, но необязательно, по меньшей мере одно из оснований в последовательности заменено аналогом основания или неосновным сайтом. В некоторых вариантах осуществления изобретения, одно или более (например, пять или менее) положений 1, 8, 9, 10, 11, 12, 14, 15, 16, 17, 18, 19 и 20 в нацеливающей области содержат дезоксирибонуклеотидное основание. В некоторых вариантах осуществления изобретения, нацеливающая область способна гибридизоваться с последовательностью, выбранной из SEQ ID NO: 51-133. В некоторых вариантах осуществления изобретения, направляющая последовательность CRISPR содержит последовательность, выбранную из SEQ ID NO: 212-231, 275-315 и 331-350. В некоторых вариантах осуществления изобретения, направляющая последовательность CRISPR содержит последовательность SEQ ID NO: 416.
[00150] В некоторых вариантах осуществления изобретения, нацеливающая область направляющей последовательности CRISPR нацелена на ген TRAC и содержит последовательность РНК GAGUCUCUCAGCUGGUACAC, где, но необязательно, по меньшей мере одно из оснований в последовательности заменено аналогом основания или неосновным сайтом. В некоторых вариантах осуществления изобретения, одно или более (например, пять или менее) положений 1, 8, 9, 10, 11, 12, 14, 15, 16, 17, 18, 19 и 20 в нацеливающей области содержат дезоксирибонуклеотидное основание. В некоторых вариантах осуществления изобретения, нацеливающая область способна гибридизоваться с последовательностью, выбранной из SEQ ID NO: 15-20. В некоторых вариантах осуществления изобретения, направляющая последовательность CRISPR содержит последовательность, выбранную из SEQ ID NO: 233-252, 317-329, 491-492 и 508. В некоторых вариантах осуществления изобретения, направляющая молекула CRISPR дополнительно содержит химическую модификацию и последовательность, выбранную из SEQ ID NO: 512-517. В некоторых вариантах осуществления изобретения, направляющая молекула CRISPR содержит последовательность SEQ ID NO: 415.
[00151] В некоторых вариантах осуществления изобретения, нацеливающая область нацелена на ген CISH и способна гибридизоваться с последовательностью, выбранной из SEQ ID NO: 157-165. В некоторых вариантах осуществления изобретения, направляющая молекула CRISPR содержит последовательность, выбранную из SEQ ID NO: 509 и 519-529.
[00152] В некоторых вариантах осуществления изобретения, нацеливающая область нацелена на ген PDCD1 и способна гибридизоваться с последовательностью, выбранной из SEQ ID NO: 135-155.
[00153] В некоторых вариантах осуществления изобретения, нацеливающая область нацелена на ген CBLB и способна гибридизоваться с последовательностью, выбранной из SEQ ID NO: 167-189. В некоторых вариантах осуществления изобретения, направляющая молекула CRISPR содержит последовательность SEQ ID NO: 510.
[00154] В некоторых своих вариантах, настоящее изобретение относится к композиции нуклеиновой кислоты/белка CRISPR, содержащей направляющую молекулу CRISPR, описанную выше, и белок Cas12. В некоторых вариантах осуществления изобретения, белок Cas12 представляет собой белок Cas12, содержащий у С-конца последовательность, которая включает линкер и сигнал локализации в ядре (NLS), и которая по меньшей мере на 80% идентична аминокислотной последовательности, выбранной из группы, состоящей из SEQ ID NO: 479-490.
[00155] В некоторых своих вариантах, настоящее изобретение относится к клетке, включающей описанную выше композицию нуклеиновой кислоты/белка CRISPR, где указанная клетка представляет собой лимфоцит, T-клетку, содержащую химерный антигенный рецептор (CAR), клетку, содержащую T-клеточный рецептор (TCR), CAR-T-клетку, сконструированную на основе TCR, опухоль-инфильтрирующий лимфоцит (TIL), CAR-TIL, дендритную клетку (DC), CAR-DC, макрофаг, CAR-макрофаг (CAR-M), природную клетку-киллер (NK), индуцированную плюрипотентную стволовую клетку (iPSC), клетку, дифференцированную из iPSC, или CAR-NK-клетку.
[00155] В некоторых своих вариантах, настоящее изобретение относится к способу получения клетки, экспрессирующей химерный антигенный рецептор (CAR), где указанный способ включает: контактирование первой последовательности нуклеиновой кислоты-мишени, содержащей последовательность TRAC в клетке с нуклеопротеиновым комплексом, включающим каталитически активный белок Cas12 и первую направляющую молекулу CRISPR, имеющую нацеливающую область, способную связываться с первой последовательностью нуклеиновой кислоты-мишени; и активирующую область, способную образовывать нуклеопротеиновый комплекс с белком Cas12, где указанная направляющая молекула CRISPR содержит рибонуклеотидные основания и по меньшей мере одно дезоксирибонуклеотидное основание в активирующей области, в нацеливающей области или в обеих областях, а нуклеопротеиновый комплекс способен расщеплять первую последовательность нуклеиновой кислоты-мишени; контактирование второй последовательности нуклеиновой кислоты-мишени, содержащей последовательность В2М в той же самой клетке с нуклеопротеиновым комплексом, включающим каталитически активный белок Cas12 и вторую направляющую молекулу CRISPR, имеющую нацеливающую область, способную связываться со второй последовательностью нуклеиновой кислоты-мишени; и активирующую область, способную образовывать нуклеопротеиновый комплекс с белком Cas12, где указанная направляющая молекула CRISPR содержит рибонуклеотидные основания и по меньшей мере одно дезоксирибонуклеотидное основание в активирующей области, в нацеливающей области или в обеих областях, а нуклеопротеиновый комплекс способен расщеплять вторую последовательность нуклеиновой кислоты-мишени; получение первого донорного полинуклеотида, кодирующего CAR, содержащий scFv, трансмембранный домен, костимулирующий домен и активирующий домен, где CAR способен встраиваться в сайт расщепления первой последовательности нуклеиновой кислоты-мишени; получение второго донорного полинуклеотида, кодирующего конструкцию для слияния B2M-HLA-E, содержащую сигнал секреции В2М, сигнальную последовательность пептида HLA-G, первую линкерную последовательность, последовательность В2М, вторую линкерную последовательность, последовательность HLA-E, где конструкция для слияния В2М-HLA-E способна встраиваться в сайт расщепления во второй последовательности нуклеиновой кислоты-мишени; расщепление первой последовательности нуклеиновой кислоты-мишени и встраивание по меньшей мере части первого донорного полинуклеотида в сайт расщепления; и расщепление второй последовательности нуклеиновой кислоты-мишени и встравивание по меньшей мере части второго донорного полинуклеотида в сайт расщепления. В некоторых вариантах осуществления изобретения, второй донорный полинуклеотид дополнительно содержит последовательность Р2А у 5'-конца конструкции для слияния B2M-HLA-E. В некоторых вариантах осуществления изобретения, первый донорный полинуклеотид содержит SEQ ID NO:413. В некоторых вариантах осуществления изобретения, второй донорный полинуклеотид содержит SEQ ID NO: 414.
[00157] В некоторых вариантах осуществления изобретения, scFv в CAR способен связываться с клеточной мишенью, выбранной из группы, состоящей из CD37, CD38, CD47, CD73, CD4, CS1, PD-L1, NGFR, ENPP3, PSCA, CD79B, TACI, VEGFR2, B7-H3, B7-H6, антигена созревания В-клеток (BCMA), CD123, CD138, CD171/L1CAM, CD19, CD20, CD22, CD30, CD33, CD70, CD371, СЕА, клаудина 18.1, клаудина 18.2, CSPG4, EFGRvIII, EpCAM, EphA2, рецептора эпидермального фактора роста, ErbB, ErbB2 (HER2), FAP, FRα, GD2, GD3, глипикана 3, IL-11Rα, IL-13Rα2, рецептора альфа IL13, антигена Льюиса Y/LeY, мезотелина, MUC1, MUC16, лигандов NKG2D, PD1, PSMA, ROR-1, SLAMF7, TAG72, ULBP и белков MICA/B, VEGF2 и WT1. В некоторых вариантах осуществления изобретения, scFv способен связываться с ВСМА и содержит первую вариабельную область, включающую аминокислотную последовательность SEQ ID NO: 474, вторую вариабельную область, включающую аминокислотную последовательность SEQ ID NO: 475, и линкер, расположенный между первой и второй вариабельными областями и содержащий аминокислотную последовательность SEQ ID NO: 476. В некоторых вариантах осуществления изобретения, scFv содержит аминокислотную последовательность SEQ ID NO: 477.
[00158] В некоторых вариантах осуществления изобретения, трансмембранный домен CAR происходит от α-цепи Т-клеточного рецептора, β-цепи Т-клеточного рецептора, цепи CD3ζ, CD28, CD3ε, CD45, CD4, CD5, CD8, CD9, CD16, CD22, CD33, CD37, CD64, CD80, CD86, CD134, CD137, ICOS, CD154 или GITR. В некоторых вариантах осуществления изобретения, костимулирующий домен CAR происходит от CD28, 4-1BB, GITR, ICOS-1, CD27, OX-40 или DAP10. В некоторых вариантах осуществления изобретения, CAR содержит трансмембранный домен, происходящий от CD8, костимулирующего домена 4-1BB и активирующего домена CD3ζ. В некоторых вариантах осуществления изобретения, вектор, содержащий последовательность CAR, включает лидерную последовательность, имеющую последовательность нуклеиновой кислоты SEQ ID NO: 478.
[00159] В некоторых вариантах осуществления изобретения, каталитически активный белок Cas12, используемый в этом способе, содержит у С-конца последовательность, которая включает линкер и сигнал локализации в ядре (NLS), и которая по меньшей мере на 80% идентична аминокислотной последовательности, выбранной из группы, состоящей из SEQ ID NO: 479-490.
[00160] В некоторых вариантах осуществления изобретения, способ дополнительно включает контактирование третьей последовательности нуклеиновой кислоты-мишени в той же самой клетке с нуклеопротеиновым комплексом, включающим каталитически активный белок Cas12 и третью направляющую молекулу CRISPR, имеющую нацеливающую область, способную связываться с третьей последовательностью нуклеиновой кислоты-мишени; и активирующую область, способную образовывать нуклеопротеиновый комплекс с белком Cas12, где указанная направляющая молекула CRISPR содержит рибонуклеотидные основания и по меньшей мере одно дезоксирибонуклеотидное основание в активирующей области, в нацеливающей области или в обеих областях, а нуклеопротеиновый комплекс способен расщеплять третью последовательность нуклеиновой кислоты-мишени; расщепление третьей последовательности нуклеиновой кислоты-мишени и делецию одного или более нуклеотидов из третьей последовательности нуклеиновой кислоты-мишени в сайте расщепления, где третья последовательность нуклеиновой кислоты-мишени выбрана из гена PDCD, гена CISH и гена CBLB.
[00161] В некоторых вариантах осуществления изобретения, CAR-экспрессирующая клетка представляет собой аллогенную или аутологичную CAR-Т-клетку, продуцированную из Т-лимфоцита.
[00162] В некоторых своих вариантах, настоящее изобретение относится к CAR-экспрессирующей клетке, полученной вышеописанным способом, где указанная клетка выбрана из лимфоцита, CAR-T-клетки, TCR-клетки, CAR-T-клетки, сконструированной на основе TCR, TIL, CAR-TIL, дендритной клетки, CAR-DC, макрофага, CAR-M, iPSC-клетки, клетки, дифференцированной из iPSC-клетки, NK-клетки или CAR-NK-клетки.
[00163] В некоторых своих вариантах, настоящее изобретение относится к способу проведения адаптивной клеточной терапии, включающему введение индивидууму, нуждающемуся в этом, CAR-экспрессирующей клетки, описанной выше. В некоторых вариантах осуществления изобретения, адаптивная клеточная терапия включает уничтожение ВСМА-позитивных раковых клеток, например, раковых клеток множественной миеломы.
Включение посредством ссылки
[00164] Все патенты, публикации и патентные заявки, упомянутые в настоящем описании, включены в настоящее изобретение посредством ссылки, так как если бы каждый отдельный патент, публикация или патентная заявка были конкретно и отдельно включены посредством ссылки в полном объеме и во всех целях.
Краткое описание чертежей
[00165] Особенности раскрытия настоящего изобретения подробно изложены в прилагаемой формуле изобретения. Для лучшего понимания особенностей и преимуществ настоящего изобретения ниже приводится подробное описание изобретения со ссылкой на иллюстративные варианты его осуществления, в которых изложены принципы его раскрытия, и на прилагаемые чертежи. Цифры не отображаются пропорционально и не масштабируются. Расположение указателей является приблизительным.
[00165] На фиг. 1А, фиг. 1B и фиг. 1C показаны примеры направляющих РНК типа V CRISPR-Cas12a.
[00167] На фиг. 2 проиллюстрировано расщепление полинуклеотида-мишени посредством комплекса «направляющая последовательность chRDNA Cas12a/нуклеопротеин».
[00168] На фиг. 3А-3I проиллюстрированы различные канонические и неканонические нуклеотиды для использования в направляющих chRDNA Cas12.
[00169] На фиг. 4 проиллюстрировано расщепление полинуклеотида-мишени посредством комплекса «направляющая последовательность chRDNA Cas12a/нуклеопротеин».
[00170] На фиг. 5 проиллюстрирована направляющая cr-РНК Cas12a.
[00171] На Фиг. 6 проиллюстрирована направляющая chRDNA Cas12a, содержащая основания ДНК в активирующей области и последовательность связывания с мишенью.
[00172] На фиг. 7 проиллюстрирована направляющая chRDNA Cas12a, содержащая основания ДНК и химически модифицированные нуклеиновые кислоты в активирующей области и в последовательности связывания с мишенью.
[00173] На фиг. 8 проиллюстрировано образование комплекса «направляющая последовательность chRDNA Cas12a/нуклеопротеин» и связывание полинуклеотида-мишени.
[00174] На фиг. 9 проиллюстрировано продуцирование инсерций или делеций (indel) в полинуклеотиде-мишени посредством комплекса «направляющая последовательность chRDNA Cas12a/нуклеопротеин».
[00175] На фиг. 10 проиллюстрирована инсерция донорной полинуклеотидной последовательности в полинуклеотид-мишень посредством комплекса «направляющая последовательность chRDNA Cas12a/нуклеопротеин».
[00175] На фиг. 11 проиллюстрирован одноцепочечный разрыв в полинуклеотиде-мишени посредством комплекса «направляющая последовательность chRDNA Cas12a/нуклеопротеин».
[00177] На фиг. 12 проиллюстрирован тандемный одноцепочечный разрыв в полинуклеотиде-мишени посредством двух комплексов «направляющая последовательность chRDNA Cas12a/нуклеопротеин» и встраивание донорной полинуклеотидной последовательности в полинуклеотид-мишень.
[00178] На фиг. 13 проиллюстрированы усредненные нормализованные скорости редактирования комплексов «направляющая последовательность chRDNA Cas12a/нуклеопротеин» с отдельным основанием ДНК в последовательности связывания с мишенью.
[00179] На фиг. 14 проиллюстрированы нормализованные скорости редактирования комплексов «направляющая последовательность chRDNA Cas12a/нуклеопротеин» с отдельным основанием ДНК в активирующей области.
[00180] На фиг. 15А и фиг. 15B проиллюстрирован фенотипический и цитотоксический профиль CAR-T-клеток, полученных с использованием комплексов «направляющая последовательность chRDNA Cas12a/нуклеопротеин».
[00181] На фиг. 16А и фиг. 16B проиллюстрирована активность редактирования комплекса «направляющая последовательность chRDNA Cas12a/нуклеопротеин» с различными конфигурациями полипептидных линкеров и последовательности локализации в ядре (NLS).
[00182] На фиг. 17 проиллюстрирована активность редактирования комплекса «направляющая последовательность chRDNA Cas12a/нуклеопротеин» при нацеливании одновременно на несколько генов с различными конфигурациями полипептидных линкеров и последовательности локализации в ядре (NLS).
Подробное описание изобретения
[00183] Следует отметить, что используемая здесь терминология предназначена только для описания конкретных вариантов осуществления изобретения, и не рассматривается как ограничение объема изобретения. Используемые в настоящей заявке и в прилагаемой формуле изобретения артикли «а» и «an» и «the», употребляемые с существительными в формах единственного числа, могут также относиться и к существительным во множественном числе, если из контекста описания не следует иное. Так, например, термин «полинуклеотид» включает один или несколько полинуклеотидов, а термин «вектор» включает один или несколько векторов.
[00184] Если это не оговорено особо, то все технические и научные термины, используемые в настоящей заявке, имеют свои общепринятые значения, известные специалистам в области, к которой относится изобретение. Хотя в настоящем описании могут быть использованы и другие подобные или эквивалентные способы и материалы, однако, предпочтительными являются материалы и способы, описанные в настоящей заявке.
[00185] Термины «SITE-Seq®» и «анализ SITE-Seq®» относятся к биохимическому методу идентификации последовательности сайтов разрезания в геномной ДНК, полученной с использованием Cas9-программированных одноцепочечных направляющих РНК (оцрРНК). Этот анализ подробно описан в публикации Cameron P. et al. (2017) Mapping the genomic landscape of CRISPR-Cas9 cleavage. Nature Methods. 14(6), 600-606. https//doi.org/10.1038/nmeth.4284).
[00185] Исходя из описания настоящего изобретения, специалист в данной области может применить обычные методы иммунологии, биохимии, химии, молекулярной биологии, микробиологии, биологии клетки, геномики и рекомбинантных полинуклеотидов, описанные, например, в следующих стандартных руководствах: Abbas et al. (Cellular and Molecular Immunology, 2017, 9th Edition, Elsevier, ISBN 978-0323479783); Butterfield et al. (Cancer Immunotherapy Principles and Practice, 2017, 1st Edition, Demos Medical, ISBN 978-1620700976); Kenneth Murphy (Janeway's Immunobiology, 2016, 9th Edition, Garland Science, ISBN 978-0815345053); Stevens et al. (Clinical Immunology and Serology: A Laboratory Perspective, 2016, 4th Edition, Davis Company, ISBN 978-0803644663); E.A. Greenfield (Antibodies: A Laboratory Manual, 2014, Second edition, Cold Spring Harbor Laboratory Press, ISBN 978-1-936113-81-1); R.I. Freshney (Culture of Animal Cells: A Manual of Basic Technique and Specialized Applications, 2016, 7th Edition, Wiley-Blackwell, ISBN 978-1118873656); C.A. Pinkert (Transgenic Animal Technology, Third Edition: A Laboratory Handbook, 2014, Elsevier, ISBN 978-0124104907); H. Hedrich (The Laboratory Mouse, 2012, Second Edition, Academic Press, ISBN 978-0123820082); Behringer et al. (Manipulating the Mouse Embryo: A Laboratory Manual, 2013, Fourth Edition, Cold Spring Harbor Laboratory Press, ISBN 978-1936113019); McPherson et al. (PCR 2: A Practical Approach, 1995, IRL Press, ISBN 978-0199634248); J.M. Walker (Methods in Molecular Biology (Series), Humana Press, ISSN 1064-3745); Rio et al. (RNA: A Laboratory Manual, 2010, Cold Spring Harbor Laboratory Press, ISBN 978-0879698911); Methods in Enzymology (Series), Academic Press; Green et al. (Molecular Cloning: A Laboratory Manual, 2012, Fourth Edition, Cold Spring Harbor Laboratory Press, ISBN 978-1605500560); G.T. Hermanson (Bioconjugate Techniques, 2013, Third Edition, Academic Press, ISBN 978-0123822390).
[00187] Кластеризованные регулярно чередующиеся короткие палиндромные повторы (CRISPR) и родственные CRISPR-ассоциированные белки (Cas-белки) составляют системы CRISPR-Cas. Классификация систем CRISPR-Cas претерпела множество изменений. В публикации Макаровой и др. (Nat. Rev. Microbiol., 2020, 18:67-83) была предложена система классификации, которая учитывает сигнатурные гены cas, специфичные для отдельных типов и подтипов систем CRISPR-Cas. Эта классификация также учитывает сходство последовательностей между несколькими общими Cas-белками, филогению наиболее консервативного Cas-белка, организацию генов и структуру массива CRISPR. Этот подход обеспечил классификационную схему, которая разделяет системы CRISPR-Cas на два отдельных класса: класс 1 и класс 2.
[00188] В системах класса 2, типа V, связывание cr-РНК и мишени включает связывание с Cas12, так же как и расщепление нуклеиновой кислоты-мишени. RuvC-подобный домен нуклеазы Cas12a, например, расщепляет обе цепи нуклеиновой кислоты-мишени в шахматном порядке, что приводит к образованию выступающих 5'- концов, что в свою очередь контрастирует с тупыми концами, образующимися при расщеплении Cas9. Эти 5'-выступы могут способствовать встраиванию ДНК с применением методов гомологичной рекомбинации.
[00189] Другие белки, связанные с cr-РНК типа V, которые участвуют в связывании с мишенью и в расщеплении, включают Cas12b (прежнее название C2c1) и Cas12c (прежнее название C2c3). Белки Cas12b и Cas12c аналогичны по длине белкам CRISPR класса 2 типа II Cas9 и CRISPR класса 2 типа V Cas12a, варьирующейся от приблизительно 1100 аминокислот до приблизительно 1500 аминокислот. Белки C2c1 и C2c3 также содержат RuvC-подобные домены нуклеазы и имеют архитектуру, подобную Cas12a. Белки C2c1 похожи на белки Cas9 в том, что им требуются cr-РНК и tracr-РНК для связывания с мишенью и расщепления, но имеют оптимальную температуру расщепления 50°C. Белки C2c1 нацелены на АТ-богатый PAM, который, подобно Cas12a, представляет собой 5'-последовательность-мишень. См., например, Shmakov et al. (Моlecular Cell, 2015, 60(3):385-397).
[00190] Подтипы CRISPR типа V включают белки Cas12 и демонстрируют широкое разнообразие последовательностей и их размеров; однако, подтипы Cas12 имеют общее эволюционное происхождение от нуклеаз TnpB, кодируемых IS605-подобными транспозонами. Из-за низкого сходства последовательностей и вероятной эволюции в результате множества независимых событий рекомбинации белков Cas12, классификация белков Cas12 на их соответствующие подтипы привела к многочисленным соглашениям по наименованию. В Таблице 1 представлены классификация и названия белков Cas12 типа V, а также их приблизительный размер, требования к направляющим последовательностям, предпочтительный полинуклеотид-мишень и репрезентативный организм, от которого они происходят.
Классификация подтипов типа V
tracr-РНК
tracr-РНК
tracr-РНК
tracr-РНК
tracr-РНК
дцДНК
дцДНК
tracr-РНК
[00191] Гомологи Cas12 могут быть идентифицированы с применением методов поиска сходства последовательностей, известных специалистам в данной области. Обычно, белок Cas12 способен взаимодействовать с родственным направляющим Cas12 с образованием комплекса направляющий Cas12/нуклеопротеин, способного связываться с последовательностью нуклеиновой кислоты-мишени. В некоторых вариантах настоящего изобретения, белок Cas12 или его гомолог представляет собой белок Cas12a или его гомолог.
[00192] Белки Cas12a включают, но не ограничиваются ими, Cas12a от бактерии Parcubacteria GWC2011_GWC2_44_17 (PbCpf1), бактерии Lachnospiraceae MC2017 (Lb3 Cpf1), Butyrivibrio proteoclasticus (BpCpf1), бактерии Peregrinibacteria GW2011_GWA_33_10 (PeCpf1), Acidaminococcus spp. BV3L6 (AsCpf1), Porphyromonas macacae (PmCpf1), бактерии Lachnospiraceae ND2006 (LbCpf1), Porphyromonas crevioricanis (PcCpf1), Prevotella disiens (PdCpf1), Moraxella bovoculi 237 (MbCpf1), Smithella sp. SC_K08D17 (SsCpf1), Leptospira inadai (LiCpf1), бактерии Lachnospiraceae MA2020 (Lb2Cpf1), Franciscella novicida U112 (FnCpf1), Candidatus methanoplasma termitum (CMtCpf1) и Eubacterium eligens (EeCpf1).
[00193] В системах типа V, связывание последовательности нуклеиновой кислоты-мишени обычно включает связывание белка Cas12 и cr-РНК, как и расщепление последовательности нуклеиновой кислоты-мишени. В системах типа V, RuvC-подобный домен нуклеазы белка Cas12 последовательно расщепляет обе цепи последовательности нуклеиновой кислоты-мишени, см. Swarts et al. (Mol. Cell, 2017, 66:221-233), что приводит к образованию выступающих 5'-концов, и в свою очередь контрастирует с тупыми концами, образующимися при расщеплении белка Cas9.
[00194] Активность расщепления белка Cas12 систем типа V может быть независимой от tracr-РНК (например, типа V-A); и для некоторых систем типа V требуется только одна cr-РНК, которая имеет структуру стебель-петля, образующую внутренний дуплекс. Белок Cas12 связывается с cr-РНК специфичным для последовательности и структуры образом посредством распознавания стебля-петли и последовательностей, прилегающих к стеблю-петле, а в первую очередь, нуклеотидов 5'-спейсерной последовательности, которые гибридизуются с последовательностью нуклеиновой кислоты-мишени. Эта структура стебель-петля обычно имеет длину от 15 до 22 нуклеотидов. Замены, которые разрушают этот дуплекс стебель-петля, отменяют активность расщепления, тогда как другие замены, которые не разрушают дуплекс стебель-петля, не отменяют активность расщепления. Определенные системы типа V требуют гибридизации между cr-РНК и tracr-РНК, такие как системы типа V-F1, V-G, V-C, V-E (CasX), V-K и V-B. См., например, Yan et. al. (Science, 2019, 363(6422):88-91).
[00195] Используемые здесь термины «направляющий» и «направляющий полинуклеотид» относятся к одному или нескольким полинуклеотидам, которые образуют нуклеопротеиновый комплекс с белком Cas, где нуклеопротеиновый комплекс предпочтительно связывается с последовательностью нуклеиновой кислоты-мишени в полинуклеотиде (по сравнению с полинуклеотидом, который не содержит последовательность нуклеиновой кислоты-мишени). Такие направляющие последовательности могут содержать рибонуклеотидные основания (например, РНК), дезоксирибонуклеотидные основания (например, ДНК), комбинации рибонуклеотидных оснований и дезоксирибонуклеотидных оснований (например, РНК/ДНК), аналоги нуклеотидов, модифицированные нуклеотиды и т.п., а также синтетические, встречающиеся в природе и не встречающиеся в природе модифицированные остатки остова или связи. Известно множество таких направляющих последовательностей, таких как, но не ограничивающихся ими, одноцепочечная направляющая РНК (включая миниатюрные и усеченные одноцепочечные направляющие РНК), cr-РНК, двойные направляющие РНК, включая, но не ограничиваясь ими, молекулы cr-РНК/tracr-РНК и т.п., использование которых зависит от конкретного белка Cas. Так, например, «направляющая последовательность, связанная с CRISPR-Cas12 типа V» представляет собой направляющую последовательность, которая специфически связывается с родственным белком Cas12 с образованием нуклеопротеинового комплекса.
[00195] Используемый здесь термин «полинуклеотид CRISPR» означает полинуклеотидную последовательность, содержащую часть направляющей молекулы. В некоторых вариантах осуществления изобретения, полинуклеотид CRISPR включает нацеливающую область и/или активирующую область.
[00195] Что касается направляющей молекулы, то используемые здесь термины «спейсер», «спейсерная последовательность», «спейсерный элемент» или «нацеливающая область» относятся к полинуклеотидной последовательности, которая может специфически гибридизоваться с последовательностью нуклеиновой кислоты-мишени. Нацеливающая область взаимодействует с последовательностью нуклеиновой кислоты-мишени посредством водородной связи между комплементарными парами оснований (то есть, спаренными основаниями). Нацеливающая область связывается с выбранной последовательностью нуклеиновой кислоты-мишени. В некоторых вариантах осуществления изобретения, последовательность-мишень представляет собой последовательность в геноме клетки, либо in vitro, либо ex vivo (например, при получении CAR-T-клеток) или in vivo (например, когда композиции вводят непосредственно индивидууму). Направляющая молекула может содержать любую последовательность или состоять из любой последовательности, выбранной для нацеливания на любую последовательность-мишень. Примерами последовательности-мишени являются последовательности, которые являются уникальными в геноме-мишени. Соответственно, нацеливающая область представляет собой последовательность, связывающуюся с нуклеиновой кислотой- мишенью. Нацеливающая область определяет местоположение сайт-специфического связывания и нуклеолитического расщепления белка Cas12. Изменчивость функциональной длины для нацеливающей области известна специалистам в данной области.
[00198] Используемый здесь термин «активирующая область» относится к части полинуклеотида, способной ассоциироваться или связываться с полипептидом Cas12, таким как полипептид Cas12a.
[00199] Используемые здесь термины «неосновный», «неосновный сайт», «неосновный нуклеотид», «не-пуриновый/не-пиримидиновый сайт» и «AP-сайт» являются синонимами и относятся к сайту в нуклеотидной последовательности, где отсутствует пуриновое или пиримидиновое основание. В некоторых вариантах осуществления изобретения, неосновные сайты содержат дезоксирибозный сайт. В других вариантах осуществления изобретения, неосновные сайты содержат рибозный сайт. В дополнительных вариантах осуществления изобретения, неосновные сайты содержат модифицированный остов, такой как фосфортиоатный остов или морфолиновый остов. Неосновный сайт не может образовывать пары водородных оснований с комплементарным азотистым основанием нуклеотида ДНК или РНК, поскольку он не содержит азотистого основания.
[00200] Используемые здесь термины «аналог основания», «неканоническое основание» и «химически модифицированное основание» относится к соединению, имеющему структурное сходство с каноническим пуриновым или пиримидиновым основанием, встречающимся в ДНК или РНК. Аналог основания может содержать модифицированный сахар и/или модифицированное азотистое основание по сравнению с пуриновым или пиримидиновым основанием, встречающимся в природе в ДНК или РНК. В некоторых вариантах осуществления изобретения, основным аналогом является инозин или дезоксиинозин, такой как 2'-дезоксиинозин. В других вариантах изобретения, аналог основания представляет собой 2'-дезоксирибонуклеозид, 2'-рибонуклеозид, 2'-дезоксирибонуклеотид или 2'-рибонуклеотид, где азотистое основание включает модифицированное основание (такое как, например, ксантин, уридин, оксанин (оксанозин), 7-метилгуанозин, дигидроуридин, 5-метилцитидин, С3-спейсер, 5-метил-dC, 5-гидроксибутинил-2'-дезоксиуридин, 5-нитроиндол, 5-метилизодезоксицитозин, изодезоксигуанозин, дезоксиуридин, изодезоксицитидин, другие 0-1 пуриновые аналоги, N-6-гидроксиламинопурин, небуларин, 7-деазагипоксантин, другие 7-деазапурины и 2-метилпурины). В некоторых вариантах осуществления изобретения, аналог оснований может быть выбран из группы, состоящей из 7-деаза-2'-дезоксиинозина, 2'-аза-2'-дезоксиинозина, PNA-инозина, морфолино-инозина, LNA-инозина, фосфорамидит-инозина, 2'-О-метоксиэтилинозина и 2'-ОМе-инозина. Термин «аналог основания» также включает, например, 2'-дезоксирибонуклеозиды, 2'-рибонуклеозиды, 2'-дезоксирибонуклеотиды или 2'-рибонуклеотиды, где азотистым основанием является замещенный гипоксантин. Так, например, замещенный гипоксантин может быть заменен галогеном, таким как фтор или хлор. В некоторых вариантах осуществления изобретения, аналогом основания может быть фторинозин или хлоринозин, такой как 2-хлоринозин, 6-хлоринозин, 8-хлоринозин, 2-фторинозин, 6-фторинозин или 8-фторинозин. В других вариантах осуществления изобретения, аналогом основания является дезоксиуридин. В других вариантах осуществления изобретения, аналог основания представляет собой имитатор нуклеиновой кислоты (такой как, например, искусственные нуклеиновые кислоты и ксенонуклеиновые кислоты (XNA)).
[00200] Используемый здесь термин «гибридная направляющая РНК/ДНК CRISPR» (chRDNA) относится к направляющей молекуле полинуклеотида, содержащей нацеливающую область, где полинуклеотид содержит РНК вместе с ДНК, введенной в полинуклеотид. В приведенных здесь вариантах осуществления изобретения, компонентом cr-РНК направляющей Cas12a является chRDNA.
[00202] Используемый здесь термин «комплекс направляющая chRDNA Cas12/нуклеопротеин» относится к направляющей молекуле chRDNA, связанной с белком Cas12 с образованием нуклеопротеинового комплекса, где нуклеопротеиновый комплекс способен сайт-направленно связываться с последовательностью нуклеиновой кислоты-мишени, комплементарной последовательности, связывающейся с нуклеиновой кислоты-мишенью и присутствующей в направляющей молекуле chRDNA. Используемый здесь термин «комплекс направляющая chRDNA Cas12а/нуклеопротеин» относится к направляющей молекуле chRDNA, связанной с белком Cas12а с образованием нуклеопротеинового комплекса, где нуклеопротеиновый комплекс способен сайт-направленно связываться с последовательностью нуклеиновой кислоты-мишени, комплементарной последовательности, связывающейся с нуклеиновой кислоты-мишенью и присутствующей в направляющей молекуле chRDNA.
[00203] Используемый здесь термин «элемент стебля» или «структура стебля» относится к двум цепям нуклеиновых кислот, которые образуют двухцепочечную область («элемент стебля»). «Элемент стебель-петля» или «структура стебель-петля» относится к стеблевой структуре, где 3'-концевые последовательности одной цепи ковалентно связаны с 5'-концевыми последовательностями второй цепи посредством нуклеотидной последовательности, обычно состоящей из одноцепочечных нуклеотидов («нуклеотидная последовательность элемента стебель-петля»). В некоторых вариантах осуществления изобретения, элемент петли содержит нуклеотидную последовательность элемента петли, содержащую от приблизительно 3 до приблизительно 20 нуклеотидов, а предпочтительно от приблизительно 4 до приблизительно 10 нуклеотидов. В некоторых вариантах осуществления изобретения, нуклеотидная последовательность элемента петли представляет собой одноцепочечную нуклеотидную последовательность неспаренных оснований нуклеиновых кислот, которые не взаимодействуют посредством образования водородной связи для создания элемента стебля в нуклеотидной последовательности элемента петли. Используемый здесь термин «элемент шпильки» также относится к структурам стебель-петля. Такие структуры хорошо известны специалистам в данной области. Спаривание оснований может быть точным, однако, как известно специалистам в данной области, элемент стебля не требует точного спаривания оснований. Таким образом, элемент стебля может включать одно или несколько ошибочных спариваний оснований или неспаренных оснований. Элемент стебель-петля может дополнительно содержать структуру псевдоузла.
[00204] Используемые здесь термины «нуклеотидная последовательность линкерного элемента», «линкерная нуклеотидная последовательность» и «линкерный полинуклеотид» являются синонимами и относятся к последовательности одного или более нуклеотидов, ковалентно присоединенных к последовательности первой нуклеиновой кислоты (5'-линкерная нуклеотидная последовательность - первая последовательность нуклеиновой кислоты-3'). В некоторых вариантах осуществления изобретения, линкерная нуклеотидная последовательность соединяет две отдельные последовательности нуклеиновых кислот с образованием одного полинуклеотида (например, 5'-последовательность первой нуклеиновой кислоты - линкерная нуклеотидная последовательность -последовательность второй нуклеиновой кислоты-3'). Другими примерами линкерных последовательностей являются, но не ограничиваются ими, 5'-последовательность первой нуклеиновой кислоты - линкерная нуклеотидная последовательность-3', и 5'-линкерная нуклеотидная последовательность - первая последовательность первой нуклеиновой кислоты - линкерная нуклеотидная последовательность-3'. В некоторых вариантах осуществления изобретения, нуклеотидная последовательность линкерного элемента может представлять собой одноцепочечную нуклеотидную последовательность неспаренных оснований нуклеиновых кислот, которые не взаимодействуют друг с другом посредством образования водородной связи для создания вторичной структуры (например, структуры стебель-петля) внутри нуклеотидной последовательности линкерного элемента. В некоторых вариантах осуществления изобретения, две одноцепочечные нуклеотидные последовательности линкерного элемента могут взаимодействовать друг с другом посредством водородной связи между двумя нуклеотидными последовательностями линкерного элемента. В некоторых вариантах осуществления изобретения, нуклеотидная последовательность линкерного элемента может иметь длину от приблизительно 1 до приблизительно 50 нуклеотидов, а предпочтительно от приблизительно 1 до приблизительно 15 нуклеотидов.
[00205] Используемый здесь термин «родственный» обычно относится к белку Cas12 (например, Cas12a) и к одной или более направляющим последовательностям типа V, связанным с CRISPR-Cas12 (например, к направляющим последовательностям chRDNA Cas12), которые способны образовывать нуклеопротеиновый комплекс, способный к сайт-направленному связыванию с последовательностью нуклеиновой кислоты-мишени, комплементарной последовательности, связывающейся с нуклеиновой кислотой-мишенью и присутствующей в одной или более направляющих последовательностей.
[00205] Термины «дикого типа», «встречающийся в природе» и «немодифицированный» используются здесь для обозначения типичной (или наиболее распространенной) формы, внешнего вида, фенотипа или штамма, существующих в природе; например, типичной формы клеток, организмов, полинуклеотидов, белков, макромолекулярных комплексов, генов, РНК, ДНК или геномов в том виде, в каком они встречаются в природном источнике и могут быть выделены из него. Форма дикого типа, внешний вид, фенотип или штамм служат исходным родителем до их требуемой модификации. Таким образом, мутантные, вариантные, сконструированные, рекомбинантные и модифицированные формы не являются формами дикого типа.
[00207] Термин «выделенный», если он относится к полипептиду, означает, что указанная молекула является отдельной и была выделена из целого организма, в которым эта молекула встречается в природе или присутствует, в основном, в отсутствии других биологических макромолекул того же типа. Термин «выделенный», если он относится к полинуклеотиду, означает, что молекула нуклеиновой кислоты полностью или частично лишена последовательностей, обычно связанных с ней в природе; или он относится к последовательности в том виде, в каком она существует в природе, но имеет связанные с ней гетерологичные последовательности, или к молекуле, отделенной от хромосомы.
[00208] Используемый здесь термин «очищенный» предпочтительно означает, что одна и та же молекула присутствует по меньшей мере на 75% по массе, более предпочтительно по меньшей мере на 85% по массе, еще более предпочтительно по меньшей мере на 95% по массе, а наиболее предпочтительно по меньшей мере на 98% по массе.
[00209] Термины «сконструированный», «генетически сконструированный», «генетически модифицированный», «рекомбинантный», «модифицированный», «не встречающийся в природе» и «ненативный» указывают на модификацию генома организма или клетки человеком. Эти термины охватывают способы геномной модификации, которые включают геномное редактирование, как определено в настоящей заявке, а также методы изменения экспрессии или методы инактивации генов, ферментативную инженерию, направленную эволюцию, конструирование на основе имеющихся знаний, методы случайного мутагенеза, перетасовку генов, оптимизацию по кодонам и т.п. Методы генной инженерии известны специалистам в данной области.
[00210] Используемые здесь термины «ковалентная связь», «ковалентно присоединенный», «ковалентно связанный», «ковалентно соединенный», «ковалентное связывание» и «молекулярная связь» являются синонимами и относятся к химической связи, которая включает совместное использование электронных пар между атомами. Примеры ковалентных связей включают, но не ограничиваются ими, фосфодиэфирные связи и фосфортиоатные связи.
[00211] Используемые здесь термины «нековалентная связь», «нековалентно присоединенный», «нековалентно связанный», «нековалентно соединенный», «нековалентное взаимодействие» и «нековалентное связывание» являются синонимами и относятся к любой относительно слабой химической связи, которая не содержит общей пары электронов. Множественные нековалентные связи часто стабилизируют конформацию макромолекул и опосредуют специфические взаимодействия между молекулами. Примеры нековалентных связей включают, но не ограничиваются ими, водородные связи, ионные взаимодействия (например, Na+Cl-), ван-дер-ваальсовы взаимодействия и гидрофобные связи.
[00212] Используемые здесь термины «водородная связь», «спаривание водород-основание» и «связанный водородной связью» являются синонимами и относятся к канонической водородной связи и неканонической водородной связи, включая, но не ограничиваясь ими, «пары оснований, связанных водородными связями Уотсона-Крика» (пары оснований, связанные водородными связями W-C или водородная связь W-C); «пары оснований, связанных водородными связями Хугстена» (водородную связь Хугстена); и «пары оснований, связанных с водородом по механизму неоднозначного спаривания» (водородную связь, связывающуюся по механизму неоднозначного спаривания). Водородная связь W-С, включая обратную водородную связь W-С, относится к спариванию пуриновых-пиримидиновых оснований, таких, как аденин:тимин, гуанин:цитозин и урацил:аденин. Водородная связь Хугстена, включая обратную водородную связь Хугстена, относится к варианту спаривания оснований в нуклеиновых кислотах, при котором два азотистых основания, по одному на каждой цепи, удерживаются вместе водородными связями в главной бороздке. Эта водородная связь без W-C может позволить третьей цепи наматываться на дуплекс и образовывать трехцепочечные спирали. Водородная связь по механизму неоднозначного спаривания, включая обратную водородную связь по механизму неоднозначного спаривания, относится к спариванию между двумя нуклеотидами в молекулах РНК, которое не подчиняется правилам спаривания пары оснований Уотсона-Крика. Существует четыре основные пары неоднозначного спаривания оснований: гуанин:урацил, инозин (гипоксантин):урацил, инозин:аденин и инозин:цитозин. Известно также, что взаимодействия оснований по механизму неоднозначного спаривания происходят между инозином и тимином и инозином и гуанином. Инозиновые основания и дезоксиинозиновые основания можно назвать «универсальными спаривающимися основаниями», поскольку они способны образовывать водородные связи с каноническими основаниями ДНК и РНК. См., например, Watkins et al. (Nucleic Acid Research, 2005, 33(19):6258-67). Правила канонического водородного связывания и неканонического водородного связывания известны специалистам в данной области. См., например, R. F. Gesteland (The RNA World, Third Edition (Cold Spring Harbor Monograph Series), 2005, Cold Spring Harbor Laboratory Press, ISBN 978-0879697396); R. F. Gesteland (The RNA World, Second Edition (Cold Spring Harbor Monograph Series), 1999, Cold Spring Harbor Laboratory Press, ISBN 978-0879695613); R. F. Gesteland (The RNA World, First Edition (Cold Spring Harbor Monograph Series), 1993, Cold Spring Harbor Laboratory Press, 978-0879694562) (см, например, Appendix 1: Structures of Base Pairs Involving at Least Two Hydrogen Bonds, I. Tinoco); W. Saenger (Principles of Nucleic Acid Structure, 1988, Springer International Publishing AG, ISBN 978-0-387-90761-1); S. Neidle (Principles of Nucleic Acid Structure, 2007, First Edition, Academic Press, ISBN 978-01236950791).
[00213] Используемые здесь термины «соединять», «соединенный» и «соединяющий» являются синонимами и относятся к ковалентной связи или нековалентной связи между двумя макромолекулами (например, полинуклеотидами, белками и т.п.).
[00214] Используемые здесь термины «последовательность нуклеиновой кислоты», «нуклеотидная последовательность» и «олигонуклеотид» являются синонимами и относятся к полимерной форме нуклеотидов. Используемый здесь термин «полинуклеотид» относится к полимерной форме нуклеотидов, которая имеет один 5'-конец и один 3'-конец и может содержать одну или несколько последовательностей нуклеиновых кислот. Нуклеотиды могут представлять собой дезоксирибонуклеотиды (ДНК), рибонуклеотиды (РНК), их аналоги или их комбинации и могут иметь любую длину. Полинуклеотиды могут выполнять любую функцию и могут иметь различные вторичные и третичные структуры. Эти термины охватывают известные аналоги природных нуклеотидов и нуклеотидов, которые модифицированы в основной, сахарной и/или фосфатной частях. Аналоги конкретного нуклеотида обладают одинаковой специфичностью спаривания оснований (например, аналог пары оснований A с T). Полинуклеотид может содержать один модифицированный нуклеотид или несколько модифицированных нуклеотидов. Примеры модифицированных нуклеотидов включают фторированные нуклеотиды, метилированные нуклеотиды, химически модифицированные сахара и аналоги нуклеотидов. Нуклеотидная структура может быть модифицирована до или после сборки полимера. После полимеризации, полинуклеотиды могут быть дополнительно модифицированы посредством, например, конъюгирования с компонентом для мечения или с компонентом, связывающимся с мишенью. Нуклеотидная последовательность может включать ненуклеотидные компоненты. Эти термины также охватывают нуклеиновые кислоты, содержащие модифицированные остатки остова или связи, которые являются синтетическими, встречающимися в природе и/или не встречающимися в природе, и имеют аналогичные свойства связывания в качестве эталонного полинуклеотида (например, ДНК или РНК). Примеры таких аналогов включают, но не ограничиваются ими, фосфортиоаты, фосфорамидиты, метилфосфонаты, хиральные метилфосфонаты, 2-О-метилрибонуклеотиды, нуклеиновые кислоты, связанные с пептидом (PNA), блокированные нуклеиновые кислоты (LNA™) (Exiqon, Woburn, MA), нуклеозиды, гликолевую нуклеиновую кислоту, мостиковые нуклеиновые кислоты и морфолиновые структуры.
[00215] Связанные с пептидом нуклеиновые кислоты (PNA) представляют собой синтетические гомологи нуклеиновых кислот, в которых полинуклеотидно-фосфатно-сахарный остов заменен гибким псевдопептидным полимером. Азотистые основания связаны с полимером. PNA обладают способностью к гибридизации с высокой аффинностью и специфичностью к комплементарным последовательностям РНК и ДНК.
[00215] В фосфоротиоатных нуклеиновых кислотах, фосфоротиоатная (PS) связь заменяет немостиковый кислород на атом серы в полинуклеотид-фосфатной цепи. Эта модификация делает межнуклеотидную связь устойчивой к разложению нуклеазой. В некоторых вариантах осуществления изобретения, фосфоротиоатные связи вводят между последними 3-5 нуклеотидами в 5'-концевых или 3'-концевых последовательностях полинуклеотидной последовательности для ингибирования разложения экзонуклеазой. Размещение фосфоротиоатных связей по всему олигонуклеотиду также способствует уменьшению степени разложения эндонуклеазами.
[00217] Треозная нуклеиновая кислота (TNA) представляет собой искусственный генетический полимер. Структура остова TNA включает повторяющиеся треозные сахара, связанные фосфодиэфирными связями. Полимеры TNA являются устойчивыми к разложению нуклеазой. TNA может подвергаться самосборке посредством образования водородных связей между парами оснований в дуплексные структуры.
[00218] Инверсии сцепления могут быть введены в полинуклеотиды с помощью «обратных фосфорамидитов» (см., например, www.ucalgary.ca/dnalab/synthesis/modifications/linkages). 3'-3'-связь на конце полинуклеотида стабилизирует полинуклеотид от разложения экзонуклеазой посредством образования олигонуклеотида, имеющего два 5'-ОН-конца, но не имеющего 3'-ОН-конца. Обычно, такие полинуклеотиды имеют фосфорамидитные группы в положении 5'-OH и диметокситритильную (DMT) защитную группу в положении 3'-OH. Обычно, защитная группа DMT находится на 5'-OH, а фосфорамидит - на 3'-OH.
[00219] Полинуклеотидные последовательности показаны здесь в обычной ориентации от 5' до 3', если это не оговорено особо.
[00220] Используемый здесь термин «идентичность последовательностей» обычно относится к проценту идентичности нуклеотидных оснований или аминокислот при сравнении первого полинуклеотида или полипептида со вторым полинуклеотидом или полипептидом, соответственно, с использованием алгоритмов, имеющих различные параметры взвешивания. Идентичность последовательностей между двумя полинуклеотидами или двумя полипептидами может быть определена с применением выравнивания последовательностей различными способами и с использованием компьютерных программ (например, BLAST, CS-BLAST, FASTA, HMMER, L-ALIGN и т.п.), доступных на web-сайтах, включая, но не ограничиваясь ими, GENBANK (www.ncbi.nlm.nih.gov/genbank/) и EMBL-EBI (www.ebi.ac.uk.). Идентичность последовательностей между двумя полинуклеотидами или двумя полипептидными последовательностями обычно вычисляют с использованием стандартных параметров по умолчанию, используемых в различных методах или компьютерных программах. Высокая степень идентичности последовательностей между двумя полинуклеотидами или двумя полипептидами обычно составляет от приблизительно 90% до 100% по длине эталонного полипептида, например, приблизительно 90% или выше, предпочтительно приблизительно 95% или выше, более предпочтительно приблизительно 98% или выше. Умеренная степень идентичности последовательностей между двумя полинуклеотидами или двумя полипептидами обычно составляет от приблизительно 80% до приблизительно 85%, например, приблизительно 80% или выше, предпочтительно приблизительно 85% по длине эталонного полипептида. Низкая степень идентичности последовательностей между двумя полинуклеотидами или двумя полипептидами обычно составляет от приблизительно 50% до 75%, например, приблизительно 50%, предпочтительно приблизительно 60%, а более предпочтительно приблизительно 75% по длине эталонного полипептида. Так, например, белок Cas12 (например, Cas12, содержащий аминокислотные замены) может иметь низкую степень идентичности последовательностей, умеренную степень идентичности последовательностей или высокую степень идентичности последовательностей по всей длине по сравнению с эталонным белком Cas12 (например, Cas12 дикого типа). В качестве другого примера, направляющая молекула может иметь низкую степень идентичности последовательностей, умеренную степень идентичности последовательностей или высокую степень идентичности последовательностей по всей длине по сравнению с эталонной направляющей молекулой дикого типа по всей длине, которая образует комплексы с эталонным белком Cas12 (например, полинуклеотидом, который образует комплекс с Cas12).
[00221] Используемый здесь термин «гибридизация», «гибридизуется» или «гибридизующийся» относится к процессу объединения двух комплементарных одноцепочечных молекул нуклеиновой кислоты (например, ДНК или РНК) с образованием одной двухцепочечной молекулы (например, ДНК/ДНК, ДНК/РНК, РНК/РНК) посредством спаривания оснований водородными связями. Жесткость гибридизации обычно определяется температурой гибридизации и концентрацией соли буфера для гибридизации; например, высокая температура и низкое содержание соли обеспечивают условия гибридизации с высокой жесткостью. Примерами диапазонов концентрации соли и температурных диапазонов для различных условий гибридизации являются: высокая жесткость, приблизительно от 0,01 до приблизительно 0,05 М соли, температура гибридизации от 5°C до 10°C ниже Tm; умеренная жесткость, приблизительно от 0,16 до приблизительно 0,33 М соли, температура гибридизации от 20°C до 29°C ниже Tm; и низкая жесткость, приблизительно от 0,33 М до приблизительно 0,82 М соли, температура гибридизации от 40°C до 48°C ниже Tm. Tm дуплексных последовательностей нуклеиновых кислот вычисляют стандартными методами, хорошо известными специалистам в данной области. См., например, Maniatis et al. (Molecular Cloning: A Laboratory Manual, 1982, Cold Spring Harbor Laboratory Press: New York); Casey et al. (Nucleic Acids Research, 1977, 4:1539-1552); Bodkin et al. (Journal of Virological Methods, 1985, 10(1):45-52); and Wallace et al. (Nucleic Acids Research, 1981, 9(4):879-894). Инструменты прогнозирования алгоритмов для оценки Tm также широко доступны. Условия высокой жесткости для гибридизации обычно относятся к условиям, при которых полинуклеотид, комплементарный последовательности-мишени, преимущественно гибридизуется с последовательностью-мишенью и, по существу, не гибридизуется с последовательностями, не являющимися мишенями. Обычно, условия гибридизации имеют умеренную жесткость, а предпочтительно высокую жесткость.
[00222] Используемый здесь термин «комплементарность» относится к способности последовательности нуклеиновой кислоты образовывать водородные связи с другой последовательностью нуклеиновой кислоты (например, посредством канонического спаривания оснований Уотсона-Крика). Процент комплементарности указывает на процент остатков в последовательности нуклеиновой кислоты, которые могут образовывать водородные связи со второй последовательностью нуклеиновой кислоты. Если две последовательности нуклеиновых кислот имеют 100% комплементарность, то эти две последовательности являются полностью комплементарными, то есть, это означает, что все смежные остатки водородной связи первого полинуклеотида комплементарны такому же количеству смежных остатков во втором полинуклеотиде.
[00223] Используемый здесь термин «соответствующее дезоксирибонуклеотидное основание», если он употребляется в отношении рибонуклеотидного основания, относится к дезоксирибонуклеотидному основанию (включая, например, модифицированный или измененный вариант канонического дезоксирибонуклеотидного основания), которое связывается посредством комплементарного спаривания оснований (Уотсона-Крика) с тем же основанием, что и рибонуклеотидное основание. Так, например, для рибонуклеотидных оснований A, C и G, соответствующими дезоксирибонуклеотидными основаниями могут быть A, C и G, соответственно. Для рибонуклеотидного основания U, соответствующим дезоксирибонуклеотидным основанием может быть, например, T.
[00224] Используемый здесь термин «связывание» относится к нековалентному взаимодействию между макромолекулами (например, между белком и полинуклеотидом, между полинуклеотидом и полинуклеотидом или между белком и белком и т.п.). Такое нековалентное взаимодействие также называют «ассоциацией» или «взаимодействием» (например, если первая макромолекула взаимодействует со второй макромолекулой, то первая макромолекула связывается со второй макромолекулой нековалентным образом). Некоторые части взаимодействия по механизму связывания могут быть последовательность-специфичными (термины «последовательность-специфическое связывание», «последовательность-специфическое взаимодействие», «сайт-специфическое связывание», и «сайт-специфическое взаимодействие» используются здесь как синонимы). Последовательность-специфическое связывание обычно относится к одной или нескольким направляющим молекулам, способным образовывать комплекс с белком (например, Cas12) для инициации связывания белка с последовательностью нуклеиновой кислоты (например, с последовательностью ДНК), содержащей последовательность нуклеиновой кислоты-мишени (например, последовательность ДНК-мишени), а предпочтительно со второй последовательностью нуклеиновой кислоты (например, со второй последовательностью ДНК) без последовательности, связывающейся с нуклеиновой кислотой-мишенью (например, без последовательности, связывающейся с ДНК-мишенью). Все компоненты связывающего взаимодействия не обязательно должны быть специфичными для последовательности, такими как контакты белка с фосфатными остатками в остове ДНК. Связывающие взаимодействия могут быть охарактеризованы константой диссоциации (Kd). «Аффинность связывания» относится к силе связывающего взаимодействия. Повышенная аффинность связывания коррелирует с более низким Kd.
[00225] Как описано в настоящей заявке, считается, что белок Cas12 «нацелен» на полинуклеотид, если комплекс направляющий Cas12/нуклеопротеин связывает или расщепляет полинуклеотид в последовательности нуклеиновой кислоты-мишени внутри полинуклеотида.
[00225] Используемый здесь термин «мотив, смежный с протоспейсером» или «PAM» относится к последовательностям двухцепочечных нуклеиновых кислот, содержащим последовательность распознавания, связывающуюся с белком Cas12, где аминокислоты белка Cas12 непосредственно взаимодействуют с последовательностью распознавания (например, белок Cas12a взаимодействует с PAM 5'-TTTN-3' или с PAM 5'-TTTV-3'). Последовательности PAM находятся на цепи, не являющейся мишенью, и могут представлять собой 5'- или 3'-последовательность, комплементарную мишени (например, в системах CRISPR-Cas12a, последовательность PAM 5'-TTTN-3' или PAM 5'-TTTV-3 находится на цепи, не являющейся мишенью, и представляет собой 5'- последовательность комплемента-мишени). PAM распознаются эффекторными белками Cas12 (например, белком Cas12a) до разматывания последовательности-мишени и связывания пары оснований водородными связями между последовательностью-мишенью и последовательностью, связывающейся с нуклеиновой кислотой-мишенью.
[00227] Используемые здесь термины «мишень», «последовательность-мишень», «последовательность нуклеиновой кислоты-мишени», «последовательность-мишень нуклеиновой кислоты» и «целевая последовательность» являются синонимами и относятся к последовательности нуклеиновой кислоты, которая полностью или частично комплементарна последовательности, связывающейся с нуклеиновой кислотой-мишенью полинуклеотида Cas12 (например, нацеливающей области). Обычно, последовательность связывания с нуклеиновой кислотой-мишенью выбирают так, чтобы она была на 100% комплементарна последовательности-мишени нуклеиновой кислоты, на которую направлено связывание нуклеопротеинового комплекса Cas12; однако, для ослабления связывания с последовательностью-мишенью нуклеиновой кислоты можно использовать более низкий процент комплементарности.
[00228] Если последовательность связывания с нуклеиновой кислотой-мишенью на 100% комплементарна последовательности-мишени, исключая неосновные сайты, содержащиеся в последовательности связывания с нуклеиновой кислотой-мишенью, то такая последовательность-мишень упоминается здесь как «целевая последовательность». Связывание с последовательностью-мишенью относится к связыванию комплекса направляющий Cas12/нуклеопротеин с последовательностью нуклеиновой кислоты, которая на 100% комплементарна неосновной части сайта последовательности, связывающейся с нуклеиновой кислотой-мишенью (спейсера). Если последовательность связывания с нуклеиновой кислотой-мишенью (спейсер) менее, чем на 100% комплементарна последовательности-мишени, исключая неосновные сайты, содержащиеся в последовательности, связывающейся с нуклеиновой кислотой-мишенью, то последовательность-мишень может называться «нецелевой последовательностью». Связывание с нецелевой последовательностью относится к связыванию комплекса направляющий Cas12/нуклеопротеин с последовательностями нуклеиновых кислот, которые менее, чем на 100% комплементарны неосновной части сайта последовательности, связывающейся с нуклеиновой кислотой-мишенью (спейсера). Последовательность нуклеиновой кислоты- мишени может представлять собой двухцепочечную или одноцепочечную молекулу ДНК. Последовательность-мишень может представлять собой двухцепочечную или одноцепочечную молекулу РНК. Последовательностью-мишенью может быть гибридная молекула РНК:ДНК. Последовательностью-мишень может присутствовать на противоположной цепи последовательности PAM.
[00229] Используемый здесь термин «двухцепочечный разрыв» (DSB) относится к разделяемым обеим цепям двухцепочечного сегмента ДНК. В некоторых случаях, если происходит такой разрыв, то можно сказать, что одна цепь имеет «липкий конец», где обнажены нуклеотиды, а не водородные связи с нуклеотидами на другой цепи. В других случаях может иметь место «тупой конец», при котором обе цепи остаются полностью спаренными друг с другом.
[00230] Используемые здесь термины «донорный полинуклеотид», «донорный олигонуклеотид», «донорная матрица», «невирусный донор» и «невирусная матрица» являются синонимами и могут представлять собой двухцепочечный полинуклеотид (например, ДНК), одноцепочечный полинуклеотид (например, ДНК или РНК), или их комбинацию. Донорные полинуклеотиды могут содержать плечи гомологии, фланкирующие последовательность вставки (например, DSB в ДНК). Плечи гомологии с каждой стороны могут отличаться по длине. Параметры для создания и конструирования донорных полинуклеотидов хорошо известны специалистам в данной области. См., например, Ran et al. (Nature Protocols, 2013, 8(11):2281-2308); Smithies et al. (Nature, 1985, 317:230-234); Thomas et al. (Cell, 1986, 44:419-428); Wu et al. (Nature Protocols, 2008, 3:1056-1076); Singer et al. (Cell, 1982, 31:25-33); Shen et al. (Genetics, 1986, 112:441-457); Watt et al. (PNAS, 1985, 82:4768-4772); Sugawara et al. (Journal of Molecular Cell Biology, 1992, 12(2):563-575); Rubnitz et al. (Journal of Molecular Cell Biology, 1984, 4(11):2253-2258); Ayares et al. (PNAS, 1986, 83(14):5199-5203); и Liskay et al. (Genetics, 1987, 115(1):161-167). В некоторых вариантах осуществления изобретения, донорный полинуклеотид содержит химерный антигенный рецептор (CAR).
[00231] Используемый здесь термин «репарация на основе гомологии» (HDR) относится к репарации ДНК, которая имеет место в клетках, например, во время репарации DSB в ДНК. HDR требует гомологии нуклеотидной последовательности и использует донорный полинуклеотид для репарации последовательности, где произошел DSB (например, в последовательности ДНК-мишени). Донорный полинуклеотид обычно имеет требуемую гомологию последовательности с последовательностью, фланкирующей DSB, а поэтому, донорный полинуклеотид может служить подходящей матрицей для репарации. HDR приводит к переносу генетической информации, например, с донорного полинуклеотида на последовательность ДНК-мишени. HDR может приводить к изменению последовательности ДНК-мишени (например, к инсерции, делеции или мутации), если донорная полинуклеотидная последовательность отличается от последовательности ДНК-мишени, и часть или весь донорный полинуклеотид включается в последовательность ДНК-мишени. В некоторых вариантах осуществления изобретения, весь донорный полинуклеотид, часть донорного полинуклеотида или копия донорного полинуклеотида интегрируются в сайт последовательности ДНК-мишени. Так, например, донорный полинуклеотид может быть использован для репарации разрыва в последовательности ДНК-мишени, где такая репарация приводит к переносу генетической информации (например, полинуклеотидных последовательностей) от донорного полинуклеотида в сайте или в непосредственной близости от разрыва в ДНК. Соответственно, новая генетическая информация (например, полинуклеотидные последовательности) может быть встроена или скопирована в последовательность ДНК-мишени.
[00232] Используемый здесь термин «независимая от гомологии интеграция мишени» (HITI) относится к репарации ДНК, которая имеет место в клетке, например, во время репарации DSB в ДНК. HITI, в отличие от HDR, не требует гомологии нуклеотидной последовательности и использует донорный полинуклеотид для репарации последовательности, где произошел DSB (например, в последовательности ДНК-мишени). HITI приводит к переносу генетической информации, например, с донорного полинуклеотида на последовательность ДНК-мишени. HITI может приводить к изменению последовательности ДНК-мишени (например, к инсерции, делеции или мутации), если донорная полинуклеотидная последовательность отличается от последовательности ДНК-мишени, и часть или весь донорный полинуклеотид включен в последовательность ДНК-мишени. В некоторых вариантах осуществления изобретения, весь донорный полинуклеотид, часть донорного полинуклеотида или копия донорного полинуклеотида интегрируются в сайт последовательности ДНК-мишени. Так, например, донорный полинуклеотид может быть использован для репарации разрыва в последовательности ДНК-мишени, где такая репарация приводит к передаче генетической информации (например, полинуклеотидных последовательностей) из донорного полинуклеотида в сайте или в непосредственной близости от разрыва в ДНК. Соответственно, новая генетическая информация (например, полинуклеотидные последовательности) может быть встроена или скопирована в последовательность ДНК-мишени.
[00233] «Геномная область» представляет собой сегмент хромосомы в геноме клетки-хозяина, который присутствует по обе стороны от сайта последовательности нуклеиновой кислоты-мишени или, альтернативно, также включает часть сайта последовательности нуклеиновой кислоты-мишени. Плечи гомологии донорного полинуклеотида имеют достаточную гомологию, чтобы подвергаться гомологичной рекомбинации с соответствующими областями генома. В некоторых вариантах осуществления изобретения, плечи гомологии донорного полинуклеотида имеют значительную гомологию последовательности с геномной областью, непосредственно фланкирующей сайт последовательности нуклеиновой кислоты-мишени; при этом, было выявлено, что плечи гомологии могут быть сконструированы так, чтобы они имели достаточную гомологию с областями генома, расположенными дальше от сайта последовательности нуклеиновой кислоты-мишени.
[00234] Используемый здесь термин «негомологичное соединение концов» (NHEJ) относится к репарации DSB в ДНК посредством прямого лигирования одного конца разрыва с другим концом разрыва без необходимости использования донорного полинуклеотида. NHEJ представляет собой путь репарации ДНК, доступный клеткам для репарации ДНК без использования матрицы для репарации. NHEJ в отсутствии донорного полинуклеотида часто приводит к случайной инсерции или удалению нуклеотидов в сайте DSB.
[00235] «Опосредованное микрогомологией соединение концов» (MMEJ) представляет собой путь репарации DSB в ДНК. MMEJ включает делеции, фланкирующие DSB, и выравнивание микрогомологичных последовательностей внутри сайта разрыва перед соединением. MMEJ определяется генетически и требует активности, например, CtIP, поли(АДФ-рибозо)полимеразы 1 (PARP1), ДНК-полимеразы тета (Pol θ), ДНК-лигазы 1 (Lig 1) или ДНК-лигазы 3 (Lig 3). Дополнительные генетические компоненты известны специалистам в данной области. См., например, Sfeir et al. (Trends in Biochemical Sciences, 2015, 40:701-714).
[00235] Используемый здесь термин «репарация ДНК» охватывает любой процесс, посредством которого клеточный механизм восстанавливает повреждение молекулы ДНК, содержащейся в клетке. Восстановленные повреждения могут включать одноцепочечные разрывы или двухцепочечные разрывы (DSB). Существует по меньшей мере три механизма восстановления DSB: HDR, NHEJ и MMEJ. «Репарация ДНК» также используется здесь для обозначения репарации ДНК, полученной в результате манипуляций человеком, где целевой локус модифицируют, например, путем инсерции, делеции или замены нуклеотидов, которые представляют собой формы редактирования генома.
[00237] Используемый здесь термин «рекомбинация» относится к процессу обмена генетической информацией между двумя полинуклеотидами.
[00238] Используемые здесь термины «регуляторные последовательности», «регуляторные элементы» и «контрольные элементы» являются синонимами и относятся к полинуклеотидным последовательностям, которые расположены выше (5'-некодирующие последовательности), внутри или ниже (3'-нетранслируемые последовательности) экспрессируемого полинуклеотида-мишени. Регуляторные последовательности влияют, например, на время транскрипции, количество или уровень транскрипции, процессинг или стабильность РНК и/или на трансляцию нуклеотидной последовательности с родственной структурой. Регуляторные последовательности могут включать последовательности связывания с активатором, энхансеры, интроны, последовательности распознавания полиаденилирования, промоторы, сайты инициации транскрипции, последовательности связывания с репрессором, структуры стебель-петля, последовательности инициации трансляции, внутренние сайты связывания с рибосомой (IRES), лидерные последовательности трансляции, последовательности терминации транскрипции (например, сигналы полиаденилирования и последовательности poly-U), последовательности терминации трансляции, сайты связывания с праймером и т.п.
[00239] Регуляторные элементы включают элементы, которые регулируют конститутивную, индуцируемую или репрессируемую экспрессию нуклеотидной последовательности в клетках-хозяевах многих= типов, и элементы, которые регулируют экспрессию нуклеотидной последовательности только в некоторых клетках-хозяевах (например, тканеспецифические регуляторные последовательности). В некоторых вариантах осуществления изобретения, вектор содержит один или несколько промоторов pol III, один или несколько промоторов pol II, один или несколько промоторов pol I или их комбинации. Примеры промоторов pol III включают, но не ограничиваются ими, промоторы U6 и H1. Примеры промоторов pol II включают, но не ограничиваются ими, промотор LTR ретровируса саркомы Рауса (RSV) (необязательно с энхансером RSV), промотор цитомегаловируса (CMV) (необязательно с энхансером CMV; см., например, Boshart et al. (Cell, 1985, 41:521-530)), промотор SV40, промотор дигидрофолатредуктазы, промотор β-актина, промотор фосфоглицеринкиназы (PGK) и промотор EF1α. Специалистам в данной области будет очевидно, что тип конструкции экспрессионного вектора может зависеть от таких факторов, как выбор клетки-хозяина, подлежащей трансформации, желаемый уровень экспрессии и т.п. Вектор может быть введен в клетки-хозяева так, чтобы это приводило к продуцированию транскриптов, белков или пептидов, включая слитые белки или пептиды, кодируемые описанными здесь последовательностями нуклеиновых кислот.
[00240] Используемый здесь термин «ген» относится к полинуклеотидной последовательности, содержащей экзоны и родственные регуляторные последовательности. Ген может дополнительно содержать интроны и/или нетранслируемые области (UTR).
[00241] Используемый здесь термин «функционально связанный» относится к полинуклеотидным последовательностям или аминокислотным последовательностям, находящимся в функциональной взаимосвязи друг с другом. Так, например, регуляторные последовательности (например, промотор или энхансер) «функционально связаны» с полинуклеотидом, кодирующим генный продукт, если регуляторные последовательности регулируют или способствуют модуляции транскрипции полинуклеотида. Функционально связанные регуляторные элементы обычно являются смежными с кодирующей последовательностью. Однако, энхансеры могут функционировать, даже если они отделены от промотора до нескольких тысяч пар оснований или более. Соответственно, некоторые регуляторные элементы могут быть функционально связаны с полинуклеотидной последовательностью, но не являются смежными с полинуклеотидной последовательностью. Аналогичным образом, элементы регуляции трансляции способствуют модуляции экспрессии белка из полинуклеотида.
[00242] Используемый здесь термин «экспрессия» относится к транскрипции полинуклеотида с ДНК-матрицы, в результате чего образуется, например, матричная РНК (мРНК) или другой РНК-транскрипт (например, некодирующий транскрипт, такой как структурные или каркасные РНК). Этот термин также относится к процессу, посредством которого транскрибируемая мРНК транслируется в пептиды, полипептиды или белки. Транскрипты и кодируемые полипептиды могут совместно называться «генными продуктами». Экспрессия может включать сплайсинг мРНК в эукариотической клетке, если полинуклеотид происходит от геномной ДНК.
[00243] «Кодирующая последовательность» или последовательность, которая «кодирует» выбранный полипептид, представляет собой молекулу нуклеиновой кислоты, которая транскрибируется (в случае ДНК) и транслируется (в случае мРНК) в полипептид in vitro или in vivo при ее помещении под контроль соответствующих регуляторных последовательностей. Границы кодирующей последовательности определяются старт-кодоном на 5'-конце и стоп-кодоном трансляции на 3'-конце. Последовательность терминации транскрипции может быть расположена на расстоянии от 3'-конца кодирующей последовательности.
[00244] Используемый здесь термин «модулировать» относится к изменению количества, степени или величины функции. Так, например, комплекс направляющий Cas12/нуклеопротеин, как описано в настоящей заявке, может модулировать активность последовательности промотора посредством связывания с последовательностью нуклеиновой кислоты-мишени в промоторе или вблизи него. В зависимости от действия, происходящего после связывания, комплекс направляющий Cas12/нуклеопротеин может индуцировать, усиливать, подавлять или ингибировать транскрипцию гена, функционально связанного с промоторной последовательностью. Таким образом, «модуляция» экспрессии генов включает как активацию генов, так и подавление генов.
[00245] Модуляция может быть проанализирована путем определения любого свойства, на которое прямо или косвенно влияет экспрессия гена-мишени. Такие свойства включают, например, изменения уровней РНК или белка, активности белка, уровней продукта, экспрессии гена или уровня активности репортерных генов. Соответственно, термины «модуляция экспрессии», «ингибирование экспрессии» и «активация экспрессии» гена могут относиться к способности комплекса направляющий Cas12/нуклеопротеин изменять, активировать или ингибировать транскрипцию гена.
[00245] Используемый здесь термин «вектор» и «плазмида» относятся к полинуклеотидному носителю для введения генетического материала в клетку. Векторы могут быть линейными или кольцевыми. Векторы могут содержать последовательность репликации, способную осуществлять репликацию вектора в подходящей клетке-хозяине (например, ориджин репликации). После трансформации подходящего хозяина, вектор может реплицироваться и функционировать независимо от генома хозяина или интегрироваться в геном хозяина. Конструкция вектора зависит, среди прочего, от предполагаемого его использования и от клетки-хозяина, и структура вектора для его конкретного использования в клетке-хозяине может быть определена специалистом в данной области. Четырьмя основными типами векторов являются плазмиды, вирусные векторы, космиды и искусственные хромосомы. Обычно, векторы содержат ориджин репликации, сайт множественного клонирования и/или селективный маркер. Вектор для экспрессии обычно содержит экспрессионный кластер. Под «рекомбинантным вирусом» подразумевается вирус, который был генетически изменен, например, путем добавления или инсерции гетерологичной конструкции нуклеиновой кислоты в вирусный геном или в его часть.
[00247] Используемый здесь термин «экспрессионный кластер» относится к полинуклеотидной конструкции, полученной с применением рекомбинантных методов или методов синтеза и содержащей регуляторные последовательности, функционально связанные с выбранным полинуклеотидом для облегчения экспрессии выбранного полинуклеотида в клетке-хозяине. Так, например, регуляторные последовательности могут способствовать транскрипции выбранного полинуклеотида в клетке-хозяине или транскрипции и трансляции выбранного полинуклеотида в клетке-хозяине. Экспрессионный кластер может быть, например, интегрирован в геном клетки-хозяина или он может присутствовать в векторе для формирования экспрессионного вектора.
[00248] Используемый здесь термин «нацеливающий вектор» означает рекомбинантную ДНК-конструкцию, обычно содержащую адаптированные цепи ДНК, гомологичные геномной ДНК, которые фланкируют элементы гена-мишени или последовательности нуклеиновой кислоты-мишени (например, DSB). Нацеливающий вектор содержит донорный полинуклеотид. Элементы гена-мишени могут быть модифицированы несколькими способами, включая делеции и/или инсерции. Дефектный ген-мишень может быть заменен функциональным геном-мишенью, или, в качестве альтернативы, функциональный ген может быть отключен. Необязательно, донорный полинуклеотид нацеливающего вектора содержит кластер для отбора, содержащий селективный маркер, который вводят в ген-мишень. Нацеливающие области, прилегающие к гену-мишени или находящиеся внутри него, могут быть использованы для воздействия на регуляцию экспрессии гена.
[00249] Используемые здесь термины «редактирование генов» или «редактирование генома» относятся к типу генной инженерии, который позволяет осуществлять генетическую модификацию, такую как инсерция, делеция или замена нуклеотидной последовательности или даже одного основания в конкретном сайте в клеточном геноме. Эти термины включают, но не ограничиваются ими, экспрессию гетерологичного гена, инсерцию или делецию гена или промотора, мутацию нуклеиновой кислоты и дизруптивную генетическую модификацию, как определено в настоящей заявке.
[00250] Используемый здесь термин «между» включает конечные значения в заданном диапазоне (например, диапазон от приблизительно 1 до приблизительно 50 нуклеотидов в длину включает диапазон от 1 нуклеотида ло 50 нуклеотидов).
[00251] Используемый здесь термин «аминокислота» относится к природным и синтетическим (неприродным) аминокислотам, включая аналоги аминокислот, модифицированные аминокислоты, пептидомиметики, глицин и оптические D- или L-изомеры.
[00252] Используемые здесь термины «пептид», «полипептид» и «белок» являются синонимами и относятся к полимерам аминокислот. Полипептид может иметь любую длину. Он может быть разветвленным или линейным, может прерываться не-аминокислотами и может содержать модифицированные аминокислоты. Эти термины также относятся к аминокислотному полимеру, который был модифицирован, например, посредством ацетилирования, образования дисульфидной связи, гликозилирования, липидизации, фосфорилирования, ПЭГилирования, биотинилирования, перекрестного сшивания и/или конъюгирования (например, с компонентом для мечения или с лигандом). Полипептидные последовательности показаны здесь в обычной ориентации от N-конца к С-концу, если это не оговорено особо. Полипептиды и полинуклеотиды могут быть получены с применением обычных методов в области молекулярной биологии. Кроме того, практически любой полипептид или полинуклеотид может быть взят из коммерчески доступных источников.
[00253] Используемые здесь термины «слитый белок» и «химерный белок» относятся к одному белку, созданному путем соединения двух или более белков, белковых доменов или фрагментов белка, которые в природе не встречаются вместе в одном белке.
[00254] Слитый белок может также содержать эпитопные метки (например, гистидиновые метки, метки FLAG® (Sigma Aldrich, St. Louis, MO), метки Myc), последовательности репортерных белков (например, глутатион-S-трансферазу, бета-галактозидазу, люциферазу, белок, флуоресцирующий в зеленом диапазоне спектра, белок, флуоресцирующий в голубом диапазоне спектра, белок, флуоресцирующий в желтом диапазоне спектра), и/или домены, связывающиеся с последовательностью нуклеиновой кислоты (например, ДНК-связывающий домен или РНК-связывающий домен). Слитый белок может содержать по меньшей мере одну последовательность локализации в ядре (NLS), такую как NLS вируса обезьян 40 (SV40) или NLS нуклеоплазмина. Слитый белок может также содержать домены-активаторы (например, факторы транскрипции теплового шока, активаторы NFKB) или домены-репрессоры (например, домен KRAB). Как описано Lupo et al. (Current Genomics, 2013, 14(4):268-278), домен KRAB представляет собой мощный модуль репрессии транскрипции и расположен в аминоконцевой последовательности большинства белков цинкового пальца C2H2. См., например, Margolin et al. (PNAS, 1994, 91:4509-4513); и Witzgall et al. (PNAS, 1994, 91:4514-4518 (1994). Домен KRAB обычно связывается с корепрессорными белками и/или факторами транскрипции посредством белок-белковых взаимодействий, что приводит к репрессии транскрипции генов, с которыми связываются белки KRAB-цинковых пальцев (KRAB-ZFP). См., например, Friedman et al. (Genes & Development, 1996, 10:2067-2678). В некоторых вариантах осуществления изобретения, линкерные последовательности нуклеиновых кислот используются для соединения двух или более белков, белковых доменов или фрагментов белка.
[00255] Используемые здесь термины «последовательность локализации в ядре» (NLS) или «сигнал локализации в ядре» относятся к полипептидной последовательности белка, который преимущественно увеличивает уровень субклеточной локализации белка в ядре клетки. Последовательности NLS обычно представляют собой положительно заряженные фрагменты аминокислот, расположенные у аминоконца («N-конца»), у карбокси-конца («С-конца») или внутри белка (или в их комбинации, то есть, где одна или более NLS расположены у N-конца, и одна или более NLS расположены у С-конца). Последовательности NLS могут быть ковалентно связаны непосредственно с белком, либо они могут быть присоединены посредством линкерного полипептида. Длина линкерных последовательностей может быть оптимизирована исходя из структурных особенностей белка (например, исходя из доступности растворителя по концам, присутствия других важных функциональных пептидных последовательностей по концам и т.п.) для гарантии доступности последовательности NLS для связывания и переноса когнатного белка импортина. Кроме того, оптимальная длина линкера может быть скринирована эмпирически (см. например, Пример 11). Последовательности NLS могут быть полностью синтетическими, или они могут происходить от эндогенных или экзогенных последовательностей белка. Компьютерные программы могут быть использованы для предсказания последовательности NLS в белке (см., например, moseslab.csb.utoronto.ca/NLSStradamus/ или nls-mapper.iab.keio.ac.jp/cgi-bin/NLS_Mapper_form.cgi). Примеры последовательностей NLS приводятся в Таблице 2.
Репрезентативные последовательности ядерной локализации
[00255] Используемый здесь термин «фрагмент» относится к части молекулы. Фрагмент может представлять собой функциональную группу или часть молекулы с множеством функциональных групп (например, которые имеют общие структурные свойства). Используемые здесь термины «фрагмент» и «функциональная группа» обычно являются синонимами, однако, более конкретно, «функциональная группа» может относиться к части молекулы, которая обладает некоторыми общими химическими свойствами. Термин «фрагмент» часто используется в качестве структурного описания. В некоторых вариантах осуществления изобретения, 5'-конец, 3'-конец или 5'-конец и 3'-конец (например, ненативный 5'-конец и/или ненативный 3'-конец в первом элементе стебля) может содержать один или несколько фрагментов.
[00257] Используемые здесь термины «модифицированный белок», «мутированный белок», «вариант белка» и «сконструированный белок» обычно относятся к белку, который был модифицирован таким образом, что он содержит ненативную последовательность (то есть, модифицированный белок имеет уникальную последовательность по сравнению с немодифицированным белком).
[00258] Используемый здесь термин «трансформация» относится к введению экзогенного полинуклеотида в клетку-хозяина, независимо от способа введения. Так, например, трансформация может быть достигнута путем прямого поглощения, трансфекции, инфицирования и т.п. Экзогенный полинуклеотид может сохраняться в виде неинтегрированного вектора, например, эписомы, или, альтернативно, он может быть интегрирован в геном хозяина.
[00259] «Клетка-хозяин» представляет собой клетку, которая была трансформирована или способна к трансформации экзогенной последовательностью ДНК. Клетка-хозяин может происходить от любого организма, имеющего одну или несколько клеток. Примеры клеток-хозяев включают, но не ограничиваются ими, прокариотическую клетку, эукариотическую клетку, бактериальную клетку, клетку архебактерий, клетку одноклеточного эукариотического организма, клетку простейших, клетку растения, клетку водоросли, клетку грибов (например, дрожжевую клетку, клетка съедобного гриба), клетку животного, клетку беспозвоночного животного, клетку позвоночного животного, такую как клетка млекопитающего (например, свиньи, коровы, козы, овцы, грызуна, крысы, мыши, примата, не являющегося человеком, человека и т.п.). Кроме того, клетка-хозяин может представлять собой стволовую клетку или клетку-предшественника, или клетку иммунной системы, такую как любая из описанных здесь клеток иммунной системы. Клеткой-хозяином может быть клетка человека. Так, например, клетками-хозяевами могут быть лимфоциты или стволовые клетки, такие как гемопоэтические стволовые клетки. Лимфоциты включают Т-клетки для клеточно-опосредованного цитотоксического адаптивного иммунитета, такие как цитотоксические CD4+- и/или CD8+-Т-клетки; природные клетки-киллеры (NK), которые участвуют в развитии клеточно-опосредованного цитотоксического врожденного иммунитета; и В-клетки для гуморального адаптивного иммунитета, регулируемого антителами. В объем этого термина также входят гемопоэтические стволовые клетки, которые дают начало лимфоидным клеткам. Кроме того, CAR-T-клетки, клетки, содержащие Т-клеточные рецепторы (TCR), включая TCR-сконструированные CAR-T-клетки, опухоль- инфильтрирующие лимфоциты (TIL), CAR TIL, CAR-NK-клетки и т.п., могут быть модифицированы с применением описанных здесь методов. В некоторых вариантах осуществления изобретения, человеческая клетка находится вне человеческого тела. В некоторых вариантах осуществления изобретения, клетки тела живого организма (например, человеческого тела) модифицируют ex vivo (то есть, вне живого организма). Термин еx vivo часто относится к медицинской процедуре, при которой орган, клетки или ткань берут у живого организма (например, у человека) для лечения или терапии, а затем возвращают в этот живой организм. Термин in vivo часто относится к медицинской процедуре, при которой орган, клетки или ткань в живом организме (например, в организме человека) подвергают лечению или терапии.
[00260] Термины «субъект», «индивидуум» или «пациент» используются здесь как синонимы и относятся к любому члену типа Chordata, включая, но не ограничиваясь ими, человека и других приматов, включая приматов, не являющихся человеком, таких как макак-резус, шимпанзе и другие виды обезьян и человекообразных обезьян; сельскохозяйственных животных, таких как крупный рогатый скот, овцы, свиньи, козы и лошади; домашних млекопитающих, таких как собаки и кошки; лабораторных животных, включая кроликов, мышей, крыс и морских свинок; птиц, включая домашних птиц, диких птиц и дичь, таких как куры, индейки и другие птицы семейства куриных, утки и гуси и т.п. Этот термин не указывает на конкретный возраст или пол. Таким образом, этот термин включает взрослых, молодых и новорожденных особей, а также самцов и самок. В некоторых вариантах осуществления изобретения, клетка-хозяин происходит от индивидуума (например, лимфоциты, стволовые клетки, клетки-предшественники или тканеспецифические клетки). В некоторых вариантах осуществления изобретения, к таким индивидуумам относятся индивидуумы, не являющиеся человеком.
[00261] Термины «эффективное количество» или «терапевтически эффективное количество» композиции или средства, такого как генетически сконструированная адоптивная клетка согласно изобретению относятся к количеству композиции или средства, достаточному для обеспечения желаемого ответа. Предпочтительно, эффективное количество позволяет предотвращать, удалять или устранять один или несколько опасных побочных эффектов. Такие ответы будут зависеть от конкретного заболевания, подвергаемого лечению. Так, например, у пациента, который проходит лечение рака с использованием адоптивной клеточной терапии, желаемый ответ может включать профилактику, предотвращение или устранение одного или более из нижеописанных заболеваний, например, лечение или профилактику реакции «трансплантат против хозяина» (GvHD), реакции «хозяин против трансплантата», синдрома высвобождения цитокинов (CRS), цитокинового шторма и уменьшение онкогенных трансформаций введенных генетически модифицированных клеток. Точное требуемое количество средства, применяемого при лечении, может варьироваться от индивидуума к индивидууму, в зависимости от вида, возраста и общего состояния здоровья индивидуума, тяжести состояния, подвергаемого лечению, и конкретно используемого модифицированного лимфоцита, способа введения и т.п. Подходящее «эффективное» количество в любом индивидуальном случае может быть определено специалистом в данной области с применением обычных экспериментов.
[00262] «Лечение» или «терапия» конкретного заболевания, такого как раковое заболевание или реакция «трансплантат против хозяина» включает: профилактику заболевания, например, предотвращение развития заболевания или снижение интенсивности заболевания у индивидуума, который может быть предрасположен к развитию этого заболевания, но у которого еще не наблюдаются или не были диагностированы симптомы такого заболевания; ингибирование заболевания, например, снижение скорости его развития, остановку его развития или обратное развитие заболевания; и/или ослабление симптомов заболевания, например, уменьшение числа симптомов у индивидуума.
[00263] Направляющие Cas12
[00264] Направляющая молекула chRDNA Cas12 согласно изобретению способна образовывать нуклеопротеиновый комплекс с родственным белком Cas12, таким как белок Cas12a, где указанный комплекс способен нацеливаться на последовательность-мишень, комплементарную нацеливающей области (спейсерную последовательность).
[00265] На фиг. 1А проиллюстрирован пример направляющей молекулы BV3l6 Cas12a Acidaminococcus spp., содержащей: активирующую область (фиг. 1A, 101), включающую дуплекс стебель-петля (фиг. 1A, 102); и спейсерную последовательность (фиг.1A, 103), содержащую последовательность связывания с мишенью (фиг. 1A, 104). На фиг. 1В проиллюстрирована альтернативная направляющая молекула Cas12a, содержащая: активирующую область (фиг. 1B, 105), содержащую дуплекс стебель-петля (фиг.1B, 106); и спейсерную последовательность (фиг.1B, 107), содержащую последовательность связывания с мишенью (фиг. 1B, 108) и 3'-удлинение (фиг. 1B, 109). 3'-удлинение (фиг. 1B, 109) может быть соединено со спейсерной последовательностью (фиг. 1B, 107) посредством линкерной последовательности. На фиг. 1С проиллюстрирована альтернативна направляющая молекула Cas12a, содержащая: активирующую область (фиг. 1C, 110), содержащую дуплекс стебель-петля (фиг.1C, 111) и линкерный нуклеотид (фиг. 1C, 114) и 5'-удлинение (фиг. 1C, 115); и спейсерную последовательность (фиг. 1C, 112), содержащую последовательность связывания с мишенью (фиг.1C, 113).
[00265] В направляющих молекулах Cas12 chRDNA согласно изобретению, нацеливающая область может содержать ДНК, РНК или смесь ДНК и РНК. В некоторых вариантах осуществления изобретения, нацеливающая область может содержать как ДНК, так и РНК. В некоторых вариантах осуществления изобретения, нацеливающая область может также содержать другие аналоги оснований, модифицированные нуклеотиды, неосновные сайты и т.п., а также синтетические, природные и не встречающиеся в природе модифицированные остатки остова или связи остова или их комбинации.
[00267] В направляющих молекулах Cas12 chRDNA согласно изобретению, активирующая область может содержать ДНК, РНК или смесь ДНК и РНК. В некоторых вариантах осуществления изобретения, активирующая область может содержать как ДНК, так и РНК. В определенных вариантах осуществления изобретения, активирующая область может также содержать другие аналоги оснований, модифицированные нуклеотиды, неосновные сайты и т.п., а также синтетические, природные и не встречающиеся в природе модифицированные остатки или связи остова или их комбинации. В некоторых вариантах осуществления изобретения, активирующая область примыкает к нацеливающей области. В некоторых вариантах осуществления изобретения, активирующая область расположена ниже нацеливающей области. В некоторых вариантах осуществления изобретения, активирующая область расположена выше нацеливающей области.
[00268] В некоторых вариантах осуществления изобретения, направляющие молекулы Cas12 chRDNA согласно изобретению содержат последовательность нуклеиновой кислоты, содержащую рибонуклеотидные основания и приблизительно 2% или менее, 3% или менее, 4% или менее, 5% или менее, 6% или менее, 7% или менее, 8% или менее, 9% или менее, 10% или менее, 11% или менее, 12% или менее, 13% или менее, 14% или менее, 15% или менее, 16% или менее, 17% или менее, 18% или менее, 19% или менее, 20% или менее, 21% или менее, 22% или менее, 23% или менее, 24% или менее, 25% или менее, 26% или менее, 27% или менее, 28% или менее, 29% или менее, 30% или менее, 31% или менее, 32% или менее, 33% или менее, 34% или менее, 35% или менее, 36% или менее, 37% или менее, 38% или менее, 39% или менее, 40% или менее, 41% или менее, 42% или менее, 43% или менее, 44% или менее, 45% или менее, 46% или менее, 47% или менее, 48% или менее, 49% или менее, 50% или менее, 55% или менее, 60% или менее, 65% или менее, 70% или менее, или 75% или менее дезоксирибонуклеотидных оснований или их вариантов или модифицированных производных.
[00269] Направляющие молекулы Cas12 chRDNA согласно изобретению могут иметь, например, длину 30-75 оснований, включая неосновные сайты. В некоторых вариантах осуществления изобретения, направляющая молекула Cas12 chRDNA имеет длину 30, 31, 32, 33, 34, 35, 36, 37, 38, 39, 40, 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51, 52, 53, 54, 55, 56, 57, 58, 59, 60, 61, 62, 63, 64, 65, 66, 67, 68, 69, 70, 71, 72, 73, 74 или 75 оснований, включая неосновные сайты. В некоторых вариантах осуществления изобретения, направляющая молекула Cas12 chRDNA имеет длину 40 оснований, включая неосновные сайты.
[00270] Используемый здесь термин «в процентах от общей длины» полинуклеотидной последовательности, такой как направляющая молекула Cas12 chRDNA, активирующая область или нацеливающая область, относится к общей длине полинуклеотидной последовательности, включающей, например, неосновные сайты и модифицированные и вариантные основания.
[00271] В некоторых вариантах осуществления изобретения, активирующая область имеет длину от 10 до 25 оснований, включая неосновные сайты. В некоторых вариантах осуществления изобретения, активирующая область имеет длину 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, или 25 оснований, включая неосновные сайты. В некоторых вариантах осуществления изобретения, активирующая область имеет длину 20 оснований, включая неосновные сайты.
[00272] В некоторых вариантах осуществления изобретения, нацеливающая область имеет длину от 10 до 30 оснований, включая неосновные сайты. В некоторых вариантах осуществления изобретения, нацеливающая область имеет длину 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, или 30 оснований, включая неосновные сайты. В некоторых вариантах осуществления изобретения, нацеливающая область имеет длину 20 оснований, включая неосновные сайты.
[00273] В приведенных здесь вариантах осуществления изобретения, активирующая область и/или нацеливающая область содержит рибонуклеотидные основания и одно или несколько дезоксирибонуклеотидных оснований. Активирующая и/или нацеливающая область могут также, в некоторых вариантах осуществления изобретения, содержать дополнительные модификации, включая аналоги оснований, модифицированные нуклеотиды, неосновные сайты или их комбинации. В некоторых вариантах осуществления изобретения, активирующая и/или нацеливающая область могут содержать синтетические, природные или не встречающиеся в природе модифицированные остатки или связи остова или их комбинации.
[00274] Одно или несколько дезоксирибонуклеотидных оснований могут присутствовать в любом одном или нескольких положениях в нацеливающей области. Так, например, для нацеливающей области длиной 30 оснований, включающей неосновные сайты, одно или несколько дезоксирибонуклеотидных оснований могут присутствовать в одном или нескольких положениях 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29 или 30. Для менее крупных нацеливающих областей, положения будут соответственно сокращены. Так, например, для нацеливающей области длиной 20 оснований, включающей неосновные сайты, одно или несколько дезоксирибонуклеотидных оснований могут присутствовать в одном или нескольких положениях 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, или 20.
[00275] Одна или несколько дополнительных модификаций (таких как аналоги оснований, модифицированные нуклеотиды, неосновные сайты, модифицированные остатки или связи остова или их комбинации) могут присутствовать в любом одном или нескольких положениях в нацеливающей области. Так, например, для нацеливающей области длиной 30 оснований, включающей неосновные сайты, одна или несколько дополнительных модификаций могут присутствовать в одном или нескольких положениях 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29 или 30. Для небольших нацеливающих областей, положения будут соответственно сокращены. Так, например, для нацеливающей области длиной 20 оснований, включающей неосновные сайты, одна или несколько дополнительных модификаций могут присутствовать в одном или нескольких положениях 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19 или 20.
[00275] Одно или несколько дезоксирибонуклеотидных оснований могут присутствовать в любом одном или нескольких положениях в активирующей области. Так, например, для активирующей области длиной 25 оснований, включающей неосновные сайты, одно или несколько дезоксирибонуклеотидных оснований могут присутствовать в одном или нескольких положениях 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24 или 25. Для меньших активирующих областей, положения будут соответственно уменьшены. Так, например, для активирующей области длиной 20 оснований, включающей неосновные сайты, одно или несколько дезоксирибонуклеотидных оснований могут присутствовать в одном или нескольких положениях 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19 или 20.
[00277] Одна или несколько дополнительных модификаций (таких как аналоги оснований, модифицированные нуклеотиды, неосновные сайты, модифицированные остатки остова или связи или их комбинации) могут присутствовать в любом одном или нескольких положениях в активирующей области. Так, например, для активирующей области длиной 25 оснований, включающей неосновные сайты, одна или более дополнительных модификаций могут присутствовать в одном или более положениях 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24 или 25. Для менее крупных активирующих областей, положения будут соответственно уменьшены. Так, например, для активирующей области длиной 20 оснований, включающей неосновные сайты, одна или несколько дополнительных модификаций могут присутствовать в одном или нескольких положениях 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19 или 20.
[00278] В некоторых вариантах осуществления изобретения, количество дезоксирибонуклеотидных оснований в процентах от общего размера направляющей Cas12 chRDNA, включая неосновные сайты, предпочтительно составляет 75% или менее. В некоторых вариантах осуществления изобретения, количество дезоксирибонуклеотидных оснований в процентах от общего размера направляющей Cas12 chRDNA, включая неосновные сайты, составляет 50% или менее, 45% или менее, 40% или менее, 35% или менее, 30% или менее, 25% или менее, 24% или менее, 23% или менее, 22% или менее, 21% или менее, 20% или менее, 19% или менее, 18% или менее, 17% или менее, 16% или менее, 15% или менее, 14% или менее, 13% или менее, 12% или менее, 11% или менее, 10% или менее, 9% или менее, 8% или менее, 7% или менее, 6% или менее, или 5% или менее.
[00279] В некоторых вариантах осуществления изобретения, количество дополнительных модификаций (таких как аналоги оснований, модифицированные нуклеотиды, неосновные сайты, модифицированные остатки или связи остова или их комбинации) в процентах от общего размера направляющей Cas12 chRDNA, включающей неосновные сайты, предпочтительно составляет 75% или менее. В некоторых вариантах осуществления изобретения, количество дополнительных модификаций в процентах от общего размера направляющей Cas12 chRDNA, включая неосновные сайты, составляет 50% или менее, 45% или менее, 40% или менее, 35% или менее, 30% или менее, 25% или менее, 24% или менее, 23% или менее, 22% или менее, 21% или менее, 20% или менее, 19% или менее, 18% или менее, 17% или менее, 16% или менее, 15% или менее, 14% или менее, 13% или менее, 12% или менее, 11% или менее, 10% или менее, 9% или менее, 8% или менее, 7% или менее, 6% или менее, или 5% или менее.
[00280] В некоторых вариантах осуществления изобретения, количество дезоксирибонуклеотидных оснований в процентах от общего размера нацеливающей области, включающей неосновные сайты, предпочтительно составляет 75% или менее. В некоторых вариантах осуществления изобретения, количество дезоксирибонуклеотидных оснований в процентах от общего размера нацеливающей области, включающей неосновные сайты, составляет 50% или менее, 45% или менее, 40% или менее, 35% или менее, 30% или менее, 25% или менее, 24% или менее, 23% или менее, 22% или менее, 21% или менее, 20% или менее, 19% или менее, 18% или менее, 17% или менее, 16% или менее, 15% или менее, 14% или менее, 13% или менее, 12% или менее, 11% или менее, 10% или менее, 9% или менее, 8% или менее, 7% или менее, 6% или менее, или 5% или менее.
[00281] В некоторых вариантах осуществления изобретения, количество дополнительных модификаций (таких как аналоги оснований, модифицированные нуклеотиды, неосновные сайты, модифицированные остатки остова или связи или их комбинации) в процентах от общего размера нацеливающей области, включающей неосновные сайты, предпочтительно составляет 75% или менее. В некоторых вариантах осуществления изобретения, количество дополнительных модификаций в процентах от общего размера нацеливающей области, включающей неосновные сайты, составляет 50% или менее, 45% или менее, 40% или менее, 35% или менее, 30% или менее, 25% или менее, 24% или менее, 23% или менее, 22% или менее, 21% или менее, 20% или менее, 19% или менее, 18% или менее, 17% или менее, 16% или менее, 15% или менее, 14% или менее, 13% или менее, 12% или менее, 11% или менее, 10% или менее, 9% или менее, 8% или менее, 7% или менее, 6% или менее, или 5% или менее.
[00282] В некоторых вариантах осуществления изобретения, количество дезоксирибонуклеотидных оснований в процентах от общего размера активирующей области, включающей неосновные сайты, предпочтительно составляет 75% или менее. В некоторых вариантах осуществления изобретения, количество дезоксирибонуклеотидных оснований в процентах от общего размера активирующей области, включающей неосновные сайты, составляет 50% или менее, 45% или менее, 40% или менее, 35% или менее, 30% или менее, 25% или менее, 24% или менее, 23% или менее, 22% или менее, 21% или менее, 20% или менее, 19% или менее, 18% или менее, 17% или менее, 16% или менее, 15% или менее, 14% или менее, 13% или менее, 12% или менее, 11% или менее, 10% или менее, 9% или менее, 8% или менее, 7% или менее, 6% или менее, или 5% или менее.
[00283] В некоторых вариантах осуществления изобретения, количество дополнительных модификаций (таких как аналоги оснований, модифицированные нуклеотиды, неосновные сайты, модифицированные остатки остова или связи или их комбинации) в процентах от общего размера активирующей области, включающей неосновные сайты, предпочтительно составляет 75% или менее. В некоторых вариантах осуществления изобретения, количество дополнительных модификаций в процентах от общего размера активирующей области, включающей неосновные сайты, составляет 50% или менее, 45% или менее, 40% или менее, 35% или менее, 30% или менее, 25% или менее, 24% или менее, 23% или менее, 22% или менее, 21% или менее, 20% или менее, 19% или менее, 18% или менее, 17% или менее, 16% или менее, 15% или менее, 14% или менее, 13% или менее, 12% или менее, 11% или менее, 10% или менее, 9% или менее, 8% или менее, 7% или менее, 6% или менее, или 5% или менее.
[00284] В некоторых вариантах осуществления изобретения, количество дезоксирибонуклеотидных оснований в активирующей области и/или нацеливающей области регулируют для обеспечения статистически значимого различия по сравнению, например, с соответствующей активирующей областью и/или нацеливающей областью без дезоксирибонуклеотидных оснований. В некоторых вариантах осуществления изобретения, статистически значимое различие представляет собой различие в редактировании мишени или молекулы, не являющейся мишенью.
[00285] В некоторых вариантах осуществления изобретения, активирующая область и нацеливающая область содержат одно или несколько дезоксирибонуклеотидных оснований. В некоторых вариантах осуществления изобретения, активирующая область содержит одно или более дезоксирибонуклеотидных оснований, а нацеливающая область не содержит никаких дезоксирибонуклеотидных оснований (например, содержит только РНК и/или модифицированные рибонуклеотиды). В некоторых вариантах осуществления изобретения, нацеливающая область содержит одно или более дезоксирибонуклеотидных оснований, а активирующая область не содержит никаких дезоксирибонуклеотидных оснований (например, содержит только РНК и/или модифицированные рибонуклеотиды).
[00285] В некоторых вариантах осуществления изобретения, активирующая область и нацеливающая область содержат одну или более дополнительных модификаций (таких как аналоги оснований, модифицированные нуклеотиды, неосновные сайты, модифицированные остатки остова или связи или их комбинации). В некоторых вариантах осуществления изобретения, активирующая область содержит одну или более дополнительных модификаций (таких как аналоги оснований, модифицированные нуклеотиды, неосновные сайты, модифицированные остатки остова или связи или их комбинации), а нацеливающая область не содержит никаких дополнительных модификаций (то есть, содержит только РНК или ДНК). В некоторых вариантах осуществления изобретения, нацеливающая область содержит одну или более дополнительных модификаций (таких как аналоги оснований, модифицированные нуклеотиды, неосновные сайты, модифицированные остатки остова или связи или их комбинации), а активирующая область не содержит никаких дополнительных модификаций (то есть, содержит только РНК или ДНК).
[00287] На фиг. 2 проиллюстрирован белок Cas12a (фиг.2, 206), связанный с родственной направляющей молекулой Cas12a chRDNA (фиг. 2, 204), содержащей последовательность связывания с мишенью (фиг. 2, 205). Комплекс направляющая Cas12a chRDNA/нуклеопротеин разматывает полинуклеотид-мишень, содержащий последовательность-мишень, а связывающаяся с мишенью последовательность направляющей молекулы Cas12 chRDNA (фиг. 2, 205) соединена водородными связями (фиг. 2, обозначена вертикальной линией между полинуклеотидами) с последовательностью-мишенью (фиг. 2, 207). На фиг. 2, полинуклеотид-мишень содержит цепь-мишень (фиг. 2, 201), содержащую последовательность-мишень (фиг. 2, 207), а цепь, не являющаяся мишенью (фиг. 2, 202), содержит последовательность PAM (фиг. 2, 203). Последовательность PAM (фиг. 2, 203) обычно расположена выше (то есть, в направлении 5') последовательности-мишени (фиг. 2, 207) на цепи, не являющейся мишенью (фиг.2, 202). Образование водородных связей между связывающейся с мишенью последовательностью направляющей молекулы Cas12a chRDNA (фиг. 2, 205) и последовательностью-мишенью (фиг. 2, 207) приводит к ступенчатому расщеплению (фиг. 2, 208) цепи-мишени (фиг.2, 201) и цепи, не являющейся мишенью (фиг. 2, 202).
[00288] На фиг. 3А-3I проиллюстрированы различные канонические и неканонические нуклеотиды для их использования в направляющих молекулах Cas12 chRDNA согласно изобретению. В Таблице 3 представлен ряд показателей, используемых на фиг. 3A-ФИГ. 3I.
Численные индикаторы канонических и неканонических нуклеотидов для их использования в направляющих молекулах Cas12 cr-РНК и chRDNA
[00289] На фиг. 4 проиллюстрирован белок Cas12a (фиг. 4, 406), связанный с родственной направляющей молекулой Cas12a chRDNA (фиг. 4, 404), содержащей последовательность связывания с мишенью (фиг. 4, 405), где последовательность связывания с мишенью (фиг. 4, 405) содержит нуклеотиды, не являющиеся нуклеотидами РНК (фиг. 4, 409), такие, как канонические и неканонические нуклеотиды, представленные на фиг. 3В-3I. Комплекс «направляющая Cas12a chRDNA/нуклеопротеин» разматывает полинуклеотид-мишень, содержащий последовательность-мишень, а связывающаяся с мишенью последовательность направляющей молекулы Cas12 chRDNA (фиг. 4, 405) соединена водородными связями (фиг. 4, обозначена вертикальной линией между полинуклеотидами) с последовательностью-мишенью (фиг. 4, 407). На фиг. 4, полинуклеотид-мишень содержит цепь-мишень (фиг. 4, 401), содержащую последовательность-мишень (фиг. 4, 407), а цепь, не являющаяся мишенью (фиг. 4, 402), содержит последовательность PAM (фиг. 4, 403). Последовательность PAM (фиг. 4, 403) обычно расположена выше (то есть, в направлении 5') последовательности-мишени (фиг. 4, 407) на цепи, не являющейся мишенью (фиг. 4, 402). Образование водородных связей между связывающейся с мишенью последовательностью направляющей молекулы Cas12a chRDNA (фиг. 4, 405) и последовательностью-мишенью (фиг. 4, 407) приводит к ступенчатому расщеплению (фиг. 4, 408) цепи-мишени (фиг. 4, 401) и цепи, не являющейся мишенью (фиг. 4, 402).
[00290] На фиг. 5 проиллюстрирован пример направляющей молекулы cr-РНК Cas12a Acidaminococcus spp. (штамм BV3L6), содержащей: активирующую область (фиг. 5, 501), содержащую дуплекс стебель-петля (фиг. 5, 502); и спейсер (фиг. 5, 503), содержащий последовательность связывания с мишенью (фиг. 5, 504). Каждое положение нуклеотида в активирующей области (фиг. 5, 501) и в спейсере (фиг. 5, 503) помечено, начиная с 5'-конца направляющей молекулы, где активирующая область и область связывания с мишенью содержат РНК.
[00291] На Фиг. 6 проиллюстрирован пример направляющей молекулы cr-РНК Cas12a Acidaminococcus spp. (штамм BV3L6), содержащей: активирующую область (фиг. 6, 601), содержащую дуплекс стебель-петля (фиг. 6, 602); и спейсер (фиг. 6, 603), содержащий последовательность связывания с мишенью (фиг. 6, 604). Каждое положение нуклеотида в активирующей области (фиг. 6, 601) и в спейсере (фиг. 6, 603) помечено, начиная с 5'-конца направляющей молекулы, где активирующая область содержит смесь РНК (закрашено белым) и ДНК (закрашено серым), и последовательность связывания с мишенью содержит смесь РНК (закрашено белым) и ДНК (закрашено серым).
[00292] На Фиг. 7 проиллюстрирован пример направляющей молекулы chRDNA Cas12a Acidaminococcus spp. (штамм BV3L6), содержащей: активирующую область (фиг. 7, 701), содержащую дуплекс стебель-петля (фиг. 7, 702); и спейсер (фиг. 7, 703), содержащий последовательность связывания с мишенью (фиг. 7, 704). Каждое положение нуклеотида в активирующей области (фиг. 7, 701) и в спейсере (фиг. 7, 703) помечено, начиная с 5'-конца направляющей молекулы, где активирующая область содержит смесь РНК (закрашено белым) и ДНК (закрашено серым). Направляющая молекула Cas12a chRDNA дополнительно содержит другие неканонические нуклеотиды, такие как химически модифицированный сахарный нуклеотид (фиг. 7, 705), неосновный рибонуклеотид (фиг. 7, 706), дезоксирибонуклеотид с химически модифицированным остовом (фиг. 7, 707), рибонуклеотид с химически модифицированным остовом (фиг. 7, 708), и неосновный дезоксирибонуклеотид (фиг. 7, 709).
[00293] На фиг. 8 проиллюстрировано образование комплекса направляющая Cas12a chRDNA/нуклеопротеин, где белок Cas12 (фиг. 8, 801) связывается с направляющей молекулой Cas12a chRDNA (фиг.8, 802) с образованием комплекса направляющая Cas12a chRDNA/нуклеопротеин (фиг. 8, 803). Комплекс направляющая Cas12a chRDNA/нуклеопротеин (фиг. 8, 803) связывается с полинуклеотидом-мишенью (фиг. 8, 804), где полинуклеотид-мишень содержит последовательность-мишень, комплементарную связывающейся с мишенью последовательности направляющей молекулы Cas12 chRDNA, и между связывающейся с мишенью последовательностью направляющей молекулы Cas12 chRDNA и последовательностью-мишенью образуются водородные связи (фиг.8, 805).
[00294] На фиг. 9 проиллюстрировано образование инсерции или делеции (indel) в полинуклеотиде-мишени с помощью комплекса направляющая Cas12a chRDNA/нуклеопротеин, где белок Cas12 (фиг. 9, 901), образованный в комплексе с направляющей молекулой Cas12 chRDNA (фиг.9, 902), связывается с полинуклеотидом-мишенью (фиг.9, 903), содержащим PAM (фиг. 9, 904), и полинуклеотид-мишень расщепляется (фиг. 9, 905) комплексом направляющая Cas12a chRDNA/нуклеопротеин. После нацеливания, комплекс направляющая Cas12a chRDNA/нуклеопротеин диссоциирует из полинуклеотида-мишени (фиг. 9, 906), где полинуклеотид-мишень содержит вышерасположенную цепь (то есть, в направлении 5') (фиг.9, 907) и нижерасположенную цепь (то есть, в направлении 3') (фиг. 9, 908) относительно PAM (фиг. 9, 904). Механизм репарации клеточной ДНК восстанавливает полинуклеотид-мишень посредством инсерции или делеции (фиг.9, 910) последовательности вокруг сайта расщепления в полинуклеотиде-мишени. Вышерасположенная цепь (фиг.9, 911) и нижерасположенная цепь (фиг. 9, 912) соединяются, и отредактированный полинуклеотид-мишень (фиг. 9, 914) содержит инсерции-делеции (фиг. 9, 913) в сайте расщепления, где отредактированный полинуклеотид-мишень имеет другую последовательность по сравнению с неотредактированным полинуклеотидом-мишенью. В некоторых вариантах осуществления изобретения, инсерция или делеция (indel) в полинуклеотиде-мишени, образованные с помощью комплекса направляющая Cas12a chRDNA/нуклеопротеин, наблюдаются внутри клетки.
[00295] На фиг. 10 проиллюстрировано включение донорной полинуклеотидной последовательности в полинуклеотид-мишень, где белок Cas12 (фиг. 10, 1001), образованный в виде комплекса с направляющей молекулой Cas12 chRDNA (фиг. 10, 1002), связывается с полинуклеотидом-мишенью (фиг. 10, 1003), содержащим PAM (фиг. 10, 1004), и полинуклеотид-мишень расщепляется (фиг. 10, 1005) посредством комплекса направляющая Cas12a chRDNA/нуклеопротеин. После нацеливания, комплекс направляющая Cas12a chRDNA/нуклеопротеин диссоциирует из полинуклеотида-мишени (фиг. 10, 1006), где полинуклеотид-мишень содержит вышерасположенную цепь (то есть, в направлении 5') (фиг. 10, 1007) и нижерасположенную цепь (то есть, в направлении 3') (фиг. 10, 1008) относительно PAM (фиг. 10, 1004), и где образуется донорный полинуклеотид (фиг. 10, 1009). Механизм репарации клеточной ДНК восстанавливает полинуклеотид-мишень (фиг. 10, 1010) посредством донорного полинуклеотида (фиг. 10, 1011). Полученный отредактированный полинуклеотид-мишень (фиг. 10, 1010) содержит донорную последовательность (фиг. 10, 1011) в сайте-мишени. В некоторых вариантах осуществления изобретения, включение донорной полинуклеотидной последовательности в полинуклеотид-мишень происходит внутри клетки.
[00295] На фиг. 11 проиллюстрирован одноцепочечный разрыв полинуклеотида-мишени, где белок Cas12 (фиг. 11, 1101), образующий комплекс с направляющей молекулой Cas12 chRDNA (фиг.11, 1102), содержащий основания ДНК в последовательности связывания с мишенью (фиг.11, 1106), связывается с полинуклеотидом-мишенью (фиг. 11, 1103), содержащим PAM (фиг.11, 1104), и полинуклеотид-мишень разрезается (фиг. 11, 1105) только в одной цепи полинуклеотида-мишени посредством комплекса направляющая Cas12a chRDNA/нуклеопротеин.
[00297] На фиг. 12 проиллюстрировано использование двух комплексов «направляющая Cas12a chRDNA/нуклеопротеин» с одноцепочечным разрывом для образования ступенчатого двухцепочечного разрыва в полинуклеотиде-мишени, где первый комплекс направляющая Cas12a chRDNA/нуклеопротеин связывается с вышерасположенной (то есть, в 5'-направлении) последовательностью-мишенью (фиг. 12, 1201) с образованием первого разрыва в полинуклеотиде-мишени (фиг. 12, 1202), а второй комплекс направляющая Cas12a chRDNA/нуклеопротеин связывается с нижерасположенной (то есть, в 3'-направлении) последовательностью-мишенью полинуклеотида-мишени (фиг. 12, 1203) с образованием второго разрыва в полинуклеотиде-мишени (фиг. 12, 1204). После тандемного расщепления, полинуклеотид-мишень после расщепления содержит вышерасположенную (то есть, в направлении 5') цепь (фиг. 12, 1205) и нижерасположенную (то есть, в направлении 3') цепь (фиг. 12, 1206) с 5'-выступами. После этого образуется донорный полинуклеотид, и механизм репарации клеточной ДНК восстанавливает полинуклеотид-мишень (фиг.12, 1207) посредством донорного полинуклеотида (фиг. 12, 1208). Полученный отредактированный полинуклеотид-мишень (фиг. 12, 1209) содержит донорную последовательность (фиг. 12, 1210) в тандемном сайте одноцепочечного разрыва. В некоторых вариантах осуществления изобретения, использование двух комплексов направляющая Cas12a chRDNA/нуклеопротеин с одноцепочечным разрывом для генерации ступенчатого DSB в полинуклеотиде-мишени происходит внутри клетки.
[00298] На фиг. 13 проиллюстрированы положения в последовательности связывания с мишенью направляющей молекулы CHRDNA Acidaminococcus spp. (штамм BV3L6) Cas12a, содержащей основания ДНК. Ось Y представляет нормализованный процент редактирования нескольких мишеней с ДНК (см. Пример 5) в одном положении в последовательности связывания с мишенью (величины ошибок показывают стандартное отклонение). Ось Х указывает на каждое положение (от 5' до 3') последовательности связывания с мишенью. Последовательность связывания с мишенью показана над графиком (фиг. 13, 1301) с предпочтительными положениями использования оснований ДНК (то есть, среднее нормализованное редактирование более 70%) показана серым. Также показано местоположение областей, активирующих Cas12a chRDNA (фиг. 13, 1302).
[00299] На фиг. 14 проиллюстрированы положения в активирующей области направляющей молекулы Acidaminococcus spp. (штамм BV3L6) Cas12a chRDNA, содержащей основания ДНК (см. Пример 8). Ось Y представляет нормализованный процент редактирования направляющей молекулы с ДНК в одном положении в активирующей области. Ось Х указывает на положения (от 5' до 3') в активирующей области. Активирующая область показана слева на графике (фиг. 14, 1401) с предпочтительными положениями использования оснований ДНК (то есть, более 70% среднего нормализованного редактирования), обозначенными серым. Также указано местоположение последовательности связывания с направляющей мишенью Cas12a chRDNA (фиг. 14, 1402).
[00300] На фиг. 15А и 15В проиллюстрирован проточный цитометрический анализ CAR-T-клеток, сконструированных с использованием комплексов направляющей Cas12a chRDNA/нуклеопротеин. На фиг. 15А показан процент клеток, экспрессирующих антитела против ВСМА-CAR (фиг. 15А, 1501), белок TRAC (фиг.15А, 1502) и белок В2М (фиг.15А, 1503). На оси Х показаны клетки, которые не были обработаны (фиг.15А, 1504), клетки, которые были трансфецированы комплексом направляющая Cas12a chRDNA/нуклеопротеин, нацеленными на ген TRAC и B2M (фиг.15А, 1505) и клетки, которые были трансфецированы обоими комплексами «направляющая Cas12a chRDNA/нуклеопротеин», нацеленными на оба гена TRAC и В2М, и трансдуцированы двумя вирусами, содержащими донорные ДНК, кодирующие ген антитела против ВСМА CAR и слитый ген В2М-HLA-E, соответственно (фиг. 15А, 1506). На оси Y показан процент клеток, позитивных по различным маркерам клеточной поверхности, как было определено с помощью проточной цитометрии. На фиг. 15В показаны результаты анализа на цитотоксичность in vitro, проводимого с использованием CAR-Т-клеток, содержащих антитела против ВСМА, B2M-HLA-E (показано серыми кружками) и с использованием контрольных TRAC-KO-T-клеток (показано черными кружками), содержащих ВСМА-позитивную клеточную линию-мишень. На оси Y показан процент цитолиза клеток-мишеней, а на оси Х показано используемое отношение Е:Т. Каждые исходные данные указывают на среднее для 3 лунок для совместного культивирования при каждом соотношении Е:Т.
[00301] На фиг. 16А и 16В проиллюстрирована активность редактирования клеток посредством комплекса направляющая Cas12a chRDNA/нуклеопротеин, содержащего множество линкеров и последовательность локализации в ядре (NLS). На оси Y на графиках фиг. 16А и 16В указан процент редактирования, вычисленный методом секвенирования следующего поколения. На фиг. 16А, на оси Х показана конфигурация линкера-NLS, где все исходные дпнные представлены в виде данных с повторностями. На фиг. 16В, на оси Х показана концентрация в пикомолях направляющей последовательности Cas12a и chRDNA (20:60 или 80:240 пмоль) всех четырех конструкций линкер-NLS, показанных на фиг. 16А, а также «неоптимизированной» конструкции линкер-NLS (фиг. 16В, 1613). Все исходные данные представляют данные для уникальной последовательности направляющей молекулы-мишени и представляют собой усредненные значения с тремя повторностями на каждое из исходных значений. На фиг. 17 проиллюстрирована активность редактирования клеток посредством комплекса направляющая Cas12a chRDNA/нуклеопротеин, образованного вместе с GS-SV40 (фиг. 7, 1708; SEQ ID NO: 479) и (G4S)2-NPL (фиг. 7, 1712; SEQ ID NO: 489) при совместной доставке множества направляющих молекул Cas12a в одной реакции трансфекции. На оси Y на фиг. 17 указан процент редактирования, вычисленный методом секвенирования следующего поколения. На фиг. 17, на оси Х показан ген-мишень в качестве гена TRAC (фиг. 17, 1701; SEQ ID NO: 36), гена B2M (фиг. 17, 1702; SEQ ID NO: 62), гена CISH (фиг. 17, 1703; SEQ ID NO: 158) или гена CBLB (фиг. 17, 1704; SEQ ID NO: 171). Комплексы направляющая Cas12a chRDNA/нуклеопротеин были использованы в качестве одного нацеливающего комплекса на одну реакцию трансфекции (фиг. 17, 1705 и фиг. 17, 1709), двух нацеливающих комплексов на одну реакцию трансфекции (фиг. 17, 1706 и фиг. 17, 1710) или четырех нацеливающих комплексов на одну реакцию трансфекции (фиг. 17, 1707 и фиг. 17, 1711). Каждая полоса на графике означает усредненное значение с двумя-тремя повторностями.
[00302] Специалистам хорошо известны способы конструирования конкретных направляющих молекул Cas12 chRDNA, в которые могут быть введены дезоксирибонуклеотидные основания и, необязательно, дополнительные модификации (такие как аналоги оснований, модифицированные нуклеотиды, неосновные сайты, модифицированные остатки остова или связи или их комбинации). См., например, Briner et al. (Molecular Cell, 2014, 56:333-339). Для этого, сначала идентифицируют геномную последовательность гена, на который должна быть нацелена мишень. Точная область выбранного гена для нацеливания зависит от конкретного применения. Так, например, для активации или репрессии гена-мишени, посредством, например, активации CRISPR или ингибирования CRISPR, комплексы направляющая Cas12a chRDNA/нуклеопротеин могут быть нацелены на промотор, регулирующий экспрессию представляющего интерес гена. Для генетического нокаута, направляющие молекулы Cas12 chRDNA могут быть сконструированы так, чтобы они нацеливались на 5'-конститутивно экспрессируемые экзоны для уменьшения вероятности удаления области-мишени из мРНК за счет альтернативного сплайсинга. Экзоны вблизи N-конца могут рассматриваться как мишени, поскольку мутации со сдвигом рамки увеличивают вероятность образования нефункционального белкового продукта. Альтернативно, родственные направляющие молекулы Cas12 chRDNA могут быть сконструированы для нацеливания на экзоны, которые кодируют известные основные белковые домены. В этом отношении, мутации, не связанные со сдвигом рамки, такие как инсерции или делеции, с большей вероятностью будут изменять функцию белка, если они присутствуют в белковых доменах, которые необходимы для обеспечения функции белка. Для редактирования генов с использованием HDR, последовательность-мишень должна быть поблизости от участка желаемого редактирования. В этом случае определяется местоположение, необходимое для редактирования, и поблизости выбирается последовательность-мишень.
[00303] В некоторых вариантах осуществления изобретения, направляющая молекула Cas12 chRDNA может быть сконструирована таким образом, чтобы комплекс направляющая Cas12a chRDNA/нуклеопротеин мог связываться вне сайта расщепления белка Cas12. В этом случае, нуклеиновая кислота-мишень может не взаимодействовать с комплексом направляющая Cas12a chRDNA/нуклеопротеин, и нуклеиновая кислота-мишень может быть вырезана (например, освобождена от комплекса направляющая Cas12a chRDNA/нуклеопротеин). В некоторых вариантах осуществления изобретения, направляющая молекула Cas12 chRDNA может быть сконструирована таким образом, чтобы комплекс направляющая Cas12a chRDNA/нуклеопротеин мог связываться внутри сайта расщепления белка Cas12. В этом случае, нуклеиновая кислота-мишень может взаимодействовать с комплексом направляющая Cas12a chRDNA/нуклеопротеин, и нуклеиновая кислота-мишень может быть связана (например, связана с комплексом направляющая Cas12a chRDNA/нуклеопротеин).
[00304] Направляющие молекулы Cas12 CHRDNA могут быть сконструированы таким образом, чтобы комплекс направляющая Cas12a chRDNA/нуклеопротеин мог гибридизоваться с множеством сайтов в образце нуклеиновой кислоты. С образцом нуклеиновой кислоты могут контактировать множество комплексов направляющая Cas12a chRDNA/нуклеопротеин. Множество комплексов направляющая Cas12a chRDNA/нуклеопротеин может содержать направляющие молекулы Cas12-chRDNA, предназначенные для гибридизации с одной и той же последовательностью. Множество комплексов направляющая Cas12a chRDNA/нуклеопротеин может содержать направляющие молекулы Cas12 chRDNA, предназначенные для гибридизации с различными последовательностями-мишенями.
[00305] Последовательности-мишени могут находиться в различных положениях внутри нуклеиновой кислоты-мишени. Эти положения могут содержать одинаковые или сходные последовательности нуклеиновых кислот-мишеней. Эти положения могут содержать различные последовательности нуклеиновых кислот-мишеней. Такие положения могут быть определены в соответствии с их расстоянием друг от друга. Такие положения могут находиться на расстоянии менее, чем 10 тысяч пар оснований (т.п.о.) друг от друга, менее, чем 8 т.п.о. друг от друга, менее, чем 6 т.п.о. друг от друга, менее, чем 4 т.п.о. друг от друга, менее, чем 2 т.п.о. друг от друга, менее, чем 1 т.п.о. друг от друга, менее, чем 900 нуклеотидов друг от друга, менее, чем 800 нуклеотидов друг от друга, менее, чем 700 нуклеотидов друг от друга, менее, чем 600 нуклеотидов друг от друга, менее, чем 500 нуклеотидов друг от друга, менее, чем 400 нуклеотидов друг от друга, менее, чем 300 нуклеотидов друг от друга, менее, чем 200 нуклеотидов друг от друга или менее, чем 100 нуклеотидов друг от друга.
[00305] Комплексы направляющая Cas12a chRDNA/нуклеопротеин могут расщеплять нуклеиновую кислоту-мишень, что может приводить к удалению нуклеиновой кислоты-мишени, которая может иметь длину менее, чем 10 тысяч пар оснований (т.п.о.), менее, чем 8 т.п.о., менее, чем 6 т.п.о., менее, чем 4 т.п.о., менее, чем 2 т.п.о., менее, чем 1 т.п.о., менее, чем 900 нуклеотидов, менее, чем 800 нуклеотидов, менее, чем 700 нуклеотидов, менее, чем 600 нуклеотидов, менее, чем 500 нуклеотидов, менее, чем 400 нуклеотидов, менее, чем 300 нуклеотидов, менее, чем 200 нуклеотидов или менее, чем 100 нуклеотидов.
[00307] Комплексы направляющая Cas12a chRDNA/нуклеопротеин могут быть связаны с фрагментированной нуклеиновой кислотой-мишенью, которая может иметь длину менее, чем 10 тысяч пар оснований (т.п.о.), менее, чем 8 т.п.о., менее, чем 6 т.п.о., менее, чем 4 т.п.о., менее, чем 2 т.п.о., менее, чем 1 т.п.о., менее, чем 900 нуклеотидов, менее, чем 800 нуклеотидов, менее, чем 700 нуклеотидов, менее, чем 600 нуклеотидов, менее, чем 500 нуклеотидов, менее, чем 400 нуклеотидов, менее, чем 300 нуклеотидов, менее, чем 200 нуклеотидов или менее, чем 100 нуклеотидов.
[00308] Направляющие молекулы Cas12 chRDNA согласно изобретению могут быть синтезированы in vitro известными способами, такими как химический синтез в растворе или на твердом носителе, или в некоторых случаях, они могут быть получены рекомбинантно. Может быть применена единая технология получения или синтеза или комбинация технологий получения и синтеза, в которых дезоксирибонуклеотидные основания и/или модификации могут быть введены в одно или более положений по всей длине последовательности.
[00309] В некоторых вариантах осуществления изобретения, направляющая молекула Cas12 CHRDNA, ее нацеливающая область или ее активирующая область сконструированы так, чтобы они содержали дезоксирибонуклеотидное(ые) основание(основания) (и/или модифицированное(ые) дезоксирибонуклеотидное(ые) основание (основания)) в определенных положениях по сравнению с эталонной направляющей молекулой Cas12 CHRDNA, эталонной нацеливающей областью или эталонной активирующей областью, каждая из которых состоит из рибонуклеотидных оснований, соответственно.
[00310] В некоторых вариантах осуществления изобретения, эталонная направляющая молекула Cas12a chRDNA содержит следующую последовательность РНК: UAAUUUCUACUUCUUGUAGAUGAGUCUCUCAGCUGGUACAC. Направляющие молекулы Cas12a CHRDNA согласно изобретению, сконструированные на основе этой эталонной последовательности РНК, включают направляющие молекулы Cas12 CHRDNA, имеющие одно или несколько дезоксирибонуклеотидных оснований в одном или нескольких положениях 1, 3, 7, 10, 12, 14, 15, 16, 17, 18, 19, 21, 28, 29, 30, 31, 32, 34, 35, 36, 37, 38, 39 и 40. В некоторых вариантах осуществления изобретения, 23 или менее, 22 или менее, 21 или менее, 20 или менее, 19 или менее, 18 или менее, 17 или менее, 16 или менее, 15 или менее, 14 или менее, 13 или менее, 12 или менее, 11 или менее, 10 или менее, 9 или менее, 8 или менее, 7 или менее, 6 или менее, 5 или менее, 4 или менее, 3 или менее, 2 или менее, или 1 из этих перечисленных положений являются дезоксирибонуклеотидными основаниями. В некоторых вариантах осуществления изобретения, одно, все или несколько из дезоксирибонуклеотидных оснований в нацеливающей области образуют канонические пары оснований с последовательностью-мишенью. В некоторых вариантах осуществления изобретения, по меньшей мере одно или более из дезоксирибонуклеотидных оснований в нацеливающей области не образуют каноническую пару оснований с последовательностью-мишенью.
[00311] В некоторых вариантах осуществления изобретения, эталонная направляющая молекула Cas12a chRDNA содержит следующую последовательность РНК: UAAUUUCUACUCUUGUAGAUAGUGGGGGUGAAUUCAGUGU. Направляющие молекулы Cas12a CHRDNA согласно изобретению, сконструированные на основе этой эталонной последовательности РНК, включают направляющие молекулы Cas12 CHRDNA, имеющие одно или несколько дезоксирибонуклеотидных оснований в одном или нескольких положениях 1, 3, 7, 10, 12, 14, 15, 16, 17, 18, 19, 21, 28, 29, 30, 31, 32, 34, 35, 36, 37, 38, 39 и 40. В некоторых вариантах осуществления изобретения, 23 или менее, 22 или менее, 21 или менее, 20 или менее, 19 или менее, 18 или менее, 17 или менее, 16 или менее, 15 или менее, 14 или менее, 13 или менее, 12 или менее, 11 или менее, 10 или менее, 9 или менее, 8 или менее, 7 или менее, 6 или менее, 5 или менее, 4 или менее, 3 или менее, 2 или менее или 1 из этих перечисленных положений являются дезоксирибонуклеотидными основаниями. В некоторых вариантах осуществления изобретения, одно, все или несколько из дезоксирибонуклеотидных оснований в нацеливающей области образуют канонические пары оснований с последовательностью-мишенью. В некоторых вариантах осуществления изобретения, по меньшей мере одно или более из дезоксирибонуклеотидных оснований в нацеливающей области не образуют каноническую пару оснований с последовательностью-мишенью.
[00312] В некоторых вариантах осуществления изобретения, эталонная активирующая область содержит последовательность РНК: UAAUUUCUACUCUUGUAGAU. Активирующие области, содержащие дезоксирибонуклеотидное основание согласно изобретению и сконструированные на основе этой эталонной последовательности РНК, включают активирующие области, имеющие одно или несколько дезоксирибонуклеотидных оснований в одном или нескольких положениях 1, 3, 7, 10, 12, 14, 15, 16, 17, 18, и 19. В некоторых вариантах осуществления изобретения, 11 или менее, 10 или менее, 9 или менее, 8 или менее, 7 или менее, 6 или менее, 5 или менее, 4 или менее, 3 или менее, 2 или менее или 1 из этих перечисленных положений представляют собой дезоксирибонуклеотидные основания.
[00313] В некоторых вариантах осуществления изобретения, эталонная нацеливающая область содержит последовательность РНК: GAGUCUCUCAGCUGGUACAC. Нацеливающие области, содержащие дезоксирибонуклеотидное основание согласно изобретению и сконструированные на основе этой эталонной последовательности РНК, включают нацеливающие области, имеющие одно или несколько дезоксирибонуклеотидных оснований в одном или нескольких положениях 1, 8, 9, 10, 11, 12, 14, 15, 16, 17, 18, 19 и 20. В некоторых вариантах осуществления изобретения, 12 или менее, 11 или менее, 10 или менее, 9 или менее, 8 или менее, 7 или менее, 6 или менее, 5 или менее, 4 или менее, 3 или менее, 2 или менее или 1 из этих перечисленных положений представляют собой дезоксирибонуклеотидные основания.
[00314] В некоторых вариантах осуществления изобретения, эталонная нацеливающая область содержит последовательность РНК: AGUGGGGGUGAAUUCAGUGU. Нацеливающие области, содержащие дезоксирибонуклеотидное основание согласно изобретению и сконструированные на основе этой эталонной последовательности РНК, включают нацеливающие области, имеющие одно или несколько дезоксирибонуклеотидных оснований в одном или нескольких положениях 1, 8, 9, 10, 11, 12, 14, 15, 16, 17, 18, 19 и 20. В некоторых вариантах осуществления изобретения, 12 или менее, 11 или менее, 10 или менее, 9 или менее, 8 или менее, 7 или менее, 6 или менее, 5 или менее, 4 или менее, 3 или менее, 2 или менее или 1 из этих перечисленных положений представляют собой дезоксирибонуклеотидные основания.
[00315] В некоторых вариантах осуществления изобретения, активирующая область имеет последовательность TrAArUrUrUCrUrACrUCrUTGrUrArGArU (где «r» предшествует рибонуклеотидному основанию; а отсутствие «r» перед основанием указывает на дезоксирибонуклеотидное основание).
[00315] В некоторых вариантах осуществления изобретения, нацеливающая область имеет последовательность GrArGrUrCrUrCrUrCrAGrCrUrGrGrUrArCrAC (где «r» предшествует рибонуклеотидному основанию; а отсутствие «r» перед основанием указывает на дезоксирибонуклеотидное основание).
[00317] В некоторых вариантах осуществления изобретения, нацеливающая область имеет последовательность ArGrUrGrGrGrGrGrUrGArArUrUrCrArGrUrGT (где «r» предшествует рибонуклеотидному основанию; а отсутствие «r» перед основанием указывает на дезоксирибонуклеотидное основание).
[00318] В некоторых вариантах осуществления изобретения, направляющая последовательность Cas12a chRDNA имеет последовательность TrAArUrUrUCrUrACrUCrUTGrUrArGArUGrArGrUrCrUrCrUrCrAGrCrUrGrGrUrArCrAC («r» предшествует рибонуклеотидному основанию; а отсутствие «r» перед основанием указывает на дезоксирибонуклеотидное основание).
[00319] В некоторых вариантах осуществления изобретения, направляющая последовательность Cas12a chRDNA имеет последовательность TrAArUrUrUCrUrACrUCrUTGrUrArGArUArGrUrGrGrGrGrGrUrGArArUrUrCrArGrUrGT (где «r» предшествует рибонуклеотидному основанию; а отсутствие «r» перед основанием указывает на дезоксирибонуклеотидное основание).
[00320] Белки Cas12
[00321] Белки Cas12 согласно изобретению включают, но не ограничиваются ими, белки дикого типа Cas12, происходящие от систем CRISPR-Cas типа V, модифицированные белки Cas12, варианты белков Cas12, ортологи Cas12 и их комбинации. В некоторых вариантах осуществления изобретения, белок Cas12 представляет собой белок Cas12a дикого типа, модифицированный белок Cas12a, вариант белка Cas12a, ортолог Cas12a или их комбинацию.
[00322] Белок Cas12 может быть модифицирован. Модификация может включать модификации аминокислоты. Модификации могут также изменять первичную аминокислотную последовательность и/или вторичную, третичную и/или четвертичную аминокислотную структуру. В некоторых вариантах осуществления изобретения, одну или несколько аминокислотных последовательностей белка Cas12 можно изменить без существенного влияния на структуру или функцию белка Cas12. Тип мутации может не иметь решающего значения, если изменение происходит в некоторых областях (например, в некритической области) белка. В зависимости от локализации замены, мутация может не оказывать существенного влияния на биологические свойства полученного варианта. Так, например, свойства и функции определенных вариантов Cas12 могут быть свойствами того же типа, что и у Cas12 дикого типа.
[00323] В некоторых случаях, тот факт может ли мутация значительно влиять на структуру и/или функцию белка Cas12 может быть определен путем выравнивания последовательности и/или структуры. Выравнивание последовательностей может идентифицировать области полипептида, которые являются сходными и/или несходными (например, консервативными, неконсервативными, гидрофобными, гидрофильными и т.п.). В некоторых случаях, область в представляющей интерес последовательности, которая аналогична другим последовательностям, является подходящей для модификации. В других случаях, область в представляющей интерес последовательности, которая отличается от других последовательностей, является подходящей для модификации. Так, например, выравнивание последовательностей может быть осуществлено путем поиска в базе данных, попарного выравнивания, выравнивания нескольких последовательностей, геномного анализа, поиска мотивов, сравнительного анализа и/или с использованием программ, таких как BLAST, CS-BLAST, HHPRED, psi-BLAST, LALIGN, PyMOL и SEQALN. Выравнивание структуры может быть осуществлено с помощью таких программ, как Dali, PHYRE, Chimera, COOT, O и PyMOL. Выравнивание может быть осуществлено путем поиска в базе данных, попарного выравнивания, выравнивания нескольких последовательностей, геномного анализа, поиска мотива или сравнительного анализа, или любой их комбинации.
[00324] Белки Cas12 обычно состоят из шести доменов, соответствующих доменам REC1, REC2, взаимодействующим с PAM (PI), нуклеазе (Nuc), Wedge (WED) и RuvC. См. например, Yamano et al. (Cell, 2016, 165(4):949-962). Домен WED и домен RuvC могут иметь трехкомпонентную архитектуру последовательностей, прерываемую последовательностями от других доменов. Так, например, последовательность домена Cas12a WED Acidaminococcus spp. прерывается последовательностями домена REC1, REC2 и PI. Кроме того, некоторые подтипы белков Cas12 содержат домен мостиковой спирали, который находится рядом с последовательностями домена RuvC или между ними.
[00325] Области белка Cas12 могут быть модифицированы для модуляции активности белка Cas12. Так, например, области белка Cas12a Acidaminococcus spp. (штамм BV3L6), соответствующие остаткам домена PI (598-718) и домена WED (526-597 и 719-883), могут быть модифицированы для изменения специфичности PAM. См. например, Tóth et al. (Nucleic Acid Research, 2020, 48(7):3722-3733). Область в белке Cas12a Acidaminococcus spp. (штамм BV3L6), соответствующая остаткам доменов REC1 (24-319) и REC2 (320-526), может быть модифицирована для изменения взаимодействия с мишенью и кинетики расщепления. Области доменов REC1 (226-304) и REC2 (368-435) взаимодействуют непосредственно с дистальным концом PAM последовательности связывания с мишенью и с последовательностью-мишенью и могут быть сконструированы для изменения эффективности расщепления последовательности-мишени. Области домена Nuc (1066-1261) и домена RuvC (940-956, 957-1065 и 1261-1307) могут быть модифицированы для изменения эффективности расщепления цепи-мишени, цепи, не являющейся мишенью, или цепи-мишени, или цепи, не являющейся мишенью в последовательности-мишени. Конструирование этих областей может включать введение мутаций, замену соответствующими областями из других ортологов Cas12, делеции, инсерции и т.п.
[00325] Модифицированные белки Cas12 могут быть использованы в комбинации с направляющими молекулами Cas12 chRDNA для изменения активности или специфичности белка Cas12. В некоторых случаях, белок Cas12 может быть модифицирован для обеспечения повышенной активности или специфичности при образовании комплекса с направляющей молекулой Cas12 chRDNA, где модификации Cas12 происходят в домене(доменах) REC1, REC2, RuvC, WED и/или Nuc. В некоторых случаях, белок Cas12 может быть модифицирован для обеспечения повышенной активности или специфичности при образовании комплекса с направляющей молекулой Cas12 chRDNA, где модификации Cas12a происходят в областях 226-304, 368-435, 940-956, 978-1158, 1159-1180, и 1181-1298 (нумерация приводится на основе последовательности Cas12a Acidominococus spp.).
[00327] Такие мутации могут быть вызваны сайт-направленным мутагенезом. Мутации могут включать замены, добавления, делеции или любую их комбинацию. В некоторых случаях, мутация представляет собой замену аминокислоты на аланин. В других случаях, мутация представляет собой замену одной аминокислоты на другую аминокислоту (например, глицин, серин, треонин, цистеин, валин, лейцин, изолейцин, метионин, пролин, фенилаланин, тирозин, триптофан, аспарагиновую кислоту, глутаминовую кислоту, аспарагин, глутамин, гистидин, лизин или аргинин). Мутация может представлять собой замену аминокислоты на неприродную аминокислоту (например, селенометионин). Мутация может представлять собой замену аминокислоты на аминокислотные миметики (например, фосфомиметики). Мутация может быть консервативной мутацией. Так, например, мутация может представлять собой замену аминокислоты на аминокислоты, которые по своему размеру, форме, заряду, полярности, конформации и/или ротамерам напоминают мутировавшие аминокислоты (например, замены цистеин/серин, замены лизин/аспарагин, замены гистидин/фенилаланин).
[00328] В некоторых вариантах осуществления изобретения, белок Cas12 представляет собой белок nCas12. Белок nCas12 представляет собой вариант белка Cas12, который является дефицитным по нуклеазе, и также называется «Cas12 с одноцепочечным разрывом» или «Cas12-никазой». В таких молекулах отсутствует часть эндонуклеазной активности и, следовательно, они могут «разрезать» только одну цепь нуклеиновой кислоты-мишени. См., например, Jinek et al. (Science, 2012, 337:816-821). Это может быть достигнуто, например, путем введения мутации (мутаций) в домен нуклеазы RuvC. Неограничивающие примеры таких модификаций могут включать D917A, E1006A и D1225A в домен нуклеазы RuvC белка Cas12a F. novicida. Очевидно, что мутация других каталитических остатков для снижения активности домена нуклеазы RuvC также может быть осуществлена специалистами в данной области. Полученный в результате белок nCas12 не способен расщеплять двухцепочечную ДНК, но сохраняет способность образовывать комплекс с направляющей молекулой, связываться с последовательностью ДНК-мишени и разрезать только одну цепь ДНК-мишени. Специфичность нацеливания определяется связыванием белка Cas12 с последовательностью PAM и комплементарным спариванием оснований направляющей молекулы с геномным локусом. В некоторых вариантах осуществления изобретения, белок nCas12 представляет собой белок nCas12a.
[00329] В некоторых вариантах осуществления изобретения, белок Cas12 представляет собой белок dCas12. Белок dCas12 представляет собой вариант белка Cas12, который является дезактивированным по нуклеазе и также называется «каталитически неактивным белком Cas12», «ферментативно неактивным Cas12», «каталитически мертвым Cas12» или «мертвым Cas12». Такие молекулы не обладают эндонуклеазной активностью, а поэтому могут быть использованы для регуляции генов на основе направляющей РНК. См., например, Jinek et al. (Science, 2012, 337:816-821). Мутации каталитических остатков для устранения активности домена RuvC могут быть осуществлены специалистами в данной области. Полученный в результате белок dCas12 не способен расщеплять двухцепочечную ДНК, но сохраняет способность образовывать комплексы с направляющей молекулой и связываться с последовательностью ДНК-мишени. Специфичность нацеливания определяется связыванием белка Cas12 с последовательностью PAM и комплементарным спариванием оснований направляющей молекулы с геномным локусом. В некоторых вариантах осуществления изобретения, белок dCas12 представляет собой белок dCas12a.
[00330] Некоторые подтипы белка Cas12 не обладают нуклеазной активностью либо из-за инактивации RuvC-подобного домена нуклеазы, либо из-за частично или полного отсутствия RuvC-подобного домена нуклеазы. Один такой подтип, типа V-K и связанный с ним белок Cas12k, вместо этого связаны с Tn7-подобными мобильными генетическими элементами tnsB, tnsC, tniQ. См., например, Strecker et al. (Science, 2019, 364(6448):48-53). Cas12k сохраняет способность образовывать комплексы с направляющей молекулой и связываться с последовательностью ДНК-мишени, а связанные с ней Tn7-подобные белки облегчают управляемую РНК транспозицию последовательностей ДНК. В некоторых вариантах осуществления изобретения, комплекс направляющая Cas12a chRDNA/нуклеопротеин представляет собой комплекс направляющая Cas12k chRDNA/нуклеопротеин.
[00331] Другие аминокислотные модификации могут включать аминокислоты с гликозилированными формами, агрегированные конъюгаты с другими молекулами и ковалентные конъюгаты с несвязанными химическими фрагментами (например, ПЭГилированными молекулами). Ковалентные варианты могут быть получены путем связывания функциональных групп с группами, которые находятся в аминокислотной цепи или в N- или С-концевом остатке. В некоторых случаях, мутированные сайт-направленные полипептиды могут также включать аллельные варианты и видовые варианты.
[00332] В некоторых вариантах осуществления изобретения, белок Cas12 может представлять собой слитый или химерный белок, содержащий первый домен белка Cas12 и второй домен другого белка, такого как белок Csy4. Модификация слияния с белком Cas12 может сообщать дополнительную активность модифицированному белку Cas12. Такие активности могут включать нуклеазную активность, метилтрансферазную активность, деметилазную активность, активность репарации ДНК, активность повреждения ДНК, активность дезаминирования, дисмутазную активность, активность алкилирования, активность депуринизации, активность окисления, активность образования пиримидиновых димеров, интегразную активность, транспозазную активность, рекомбиназную активность, полимеразную активность, лигазную активность, геликазную активность, фотолиазную активность, гликозилазную активность, ацетилтрансферазную активность, деацетилазную активность, киназную активность, фосфатазную активность, убихитинлигазную активность, активность обратной транскриптазы, активность деубихитинизации, активность аденилирования, активность деаденилирования, активность SUMOилирования, активность деSUMOсумоилирования, активность рибозилирования, активность дерибозилирования и/или активность миристоилирования или демиристоилирования, которые модифицируют полипептид, связанный с последовательностью нуклеиновой кислоты-мишени (например, с гистоном).
[00333] В некоторых вариантах осуществления изобретения, белок Cas12 может содержать одну или более последовательностей NLS (например, добавленных к последовательности белка Cas12 и/или встроенных в него). Последовательность NLS может быть локализована, например, у N-конца, у С-конца или внутри белка Cas12 (такого как белок Cas12а), включая их комбинации (например, одну или более NLS у N-конца и одну или более NLS у С-конца).
[00334] В некоторых вариантах осуществления изобретения, белок Cas12, включая белок Cas12а, может содержать множество последовательностей NLS, например, по меньшей мере 2, по меньшей мере 3, по меньшей мере 4 или по меньшей мере 5 последовательностей NLS. Множество последовательностей NLS может присутствовать у одного конца белка Cas12а (например, последовательности NLS присутствуют только у N-конца или только у С-конца), либо оно может присутствовать у обоих концов (например, одна или более последовательностей NLS у N-конца и одна или более последовательностей NLS у С-конца). Последовательности NLS могут быть полностью синтетическими, модифицированными или могут происходть от эндогенных или экзогенных последовательностей белка. В некоторых вариантах осуществления изобретения, белок Cas12, включая белок Cas12а, может содержать последовательность NLS или модифицированную последовательность или последовательность, происходящую от последовательности NLS, выбранной из крупного Т-антигена SV40, нуклеоплазмина, 53BP1, VACM-1/CUL5, CXCR4, VP1, ING4, IER5, ERK5, UL79, EWS, Hrp1, cMyc(1), cMyc(2), мышиного с-ABLE IV, Matα2 и MINIYO.
[00335] В некоторых вариантах осуществления изобретения, белок Cas12, включая белок Cas12а, может содержать последовательность NLS или модифицированную последовательность или последовательность, происходящую от последовательности NLS, выбранной из любой SEQ ID NO: 04, 05 и 493-507. Модифицированная последовательность NLS или ее производное может содержать, например, 5 или менее, 4 или менее, 3 или менее, 2 или менее или 1 аминокислотную замену; 5 или менее, 4 или менее, 3 или менее, 2 или менее или 1 аминокислотную делецию; и/или 5 или менее, 4 или менее, 3 или менее, 2 или менее или 1 аминокислотное добавление по сравнению с эталонной последовательностью NLS (например, последовательностью NLS, выбранной из любой из SEQ ID NO: 04, 05 и 493-507).
[00335] В некоторых вариантах осуществления изобретения, последовательность NLS может иметь, например, последовательность, которая по меньшей мере на 80%, по меньшей мере на 85%, по меньшей мере на 90%, по меньшей мере на 91%, по меньшей мере на 92%, по меньшей мере на 93%, по меньшей мере на 94%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, или по меньшей мере на 99% идентична последовательности NLS, выбранной из любой из SEQ ID NO: 04, 05 и 493-507.
[00337] Последовательности NLS могут быть ковалентно связаны (например, с белком Cas12, с другой(другими) последовательностью(ями) NLS или последовательностью слитого белка, связанной с белком Cas12) либо непосредственно, либо посредством линкерного полипептида. Длина линкерной последовательности может быть оптимизирована в зависимости от структурных особенностей конкретного белка Cas12 (например, от доступности растворителя по концам, присутствия других важных функциональных пептидных последовательностей по концам и т.п.) для обеспечения доступности последовательности NLS для связывания и переноса родственного белка импортина. Кроме того, как описано в Примере 11, может быть проведен эмпрический скрининг на нужную длину линкера.
[00338] В некоторых вариантах осуществления изобретения, последовательности NLS ковалентно связаны (например, с белком Cas12, с другой(другими) последовательностью(ями) NLS или последовательностью слитого белка, связанной с белком Cas12) посредством линкерной последовательности, содержащей одну или несколько аминокислот. В некоторых вариантах осуществления изобретения, линкерная последовательность содержит по меньшей мере один остаток глицина, серина и/или треонина. В некоторых вариантах осуществления изобретения, линкерная последовательность содержит по меньшей мере один остаток глицина и по меньшей мере один остаток серина. В некоторых вариантах осуществления изобретения, линкерная последовательность содержит множество остатков глицина и по меньшей мере один остаток серина. В некоторых вариантах осуществления изобретения, линкерная последовательность состоит из последовательности GS или содержит эту последовательность. В некоторых вариантах осуществления изобретения, линкерная последовательность состоит из последовательности GGGGS или содержит эту последовательность. В некоторых вариантах осуществления изобретения, линкерная последовательность состоит из последовательности GGGGSGGGGS или содержит эту последовательность.
[00339] В некоторых вариантах осуществления изобретения, белок Cas12а содержит по меньшей мере одну линкерную последовательность и по меньшей мере одну последовательность NLS у С-конца. В некоторых вариантах осуществления изобретения, по меньшей мере одна последовательность NLS выбрана из крупного Т-антигена SV40 и нуклеоплазмина или из последовательностей, которые были модифицированы или которые происходят от этих последовательностей.
[00340] В некоторых вариантах осуществления изобретения, белок Cas12а содержит линкерную последовательность GGGGSGGGGS и последовательность NLS нуклеоплазмина у С-конца, где последовательность NLS нуклеоплазмина расположена с С-концевой стороны линкерной последовательности GGGGSGGGGS.
[00341] В некоторых вариантах осуществления изобретения, белок Cas12а содержит по меньшей мере одну линкерную последовательность GS, последовательность NLS крупного Т-антигена SV40 и последовательность NLS нуклеоплазмина у С-конца, где последовательность NLS нуклеоплазмина расположена с С-концевой стороны последовательности NLS крупного Т-антигена SV40. В некоторых вариантах осуществления изобретения, первая линкерная последовательность GS присутствует с N-концевой стороны последовательности NLS крупного Т-антигена SV40, а вторая линкерная последовательность GS присутствует между последовательностью NLS крупного Т-антигена SV40 и последовательностью NLS нуклеоплазмина.
[00342] В некоторых вариантах осуществления изобретения, белок Cas12а содержит одну линкерную последовательность GS, линкерную последовательность GGGGSGGGGS, последовательность NLS крупного Т-антигена SV40 и последовательность NLS нуклеоплазмина у С-конца, где последовательность NLS нуклеоплазмина расположена с С-концевой стороны последовательности NLS крупного Т-антигена SV40. В некоторых вариантах осуществления изобретения, линкерная последовательность GGGGSGGGGS присутствует с N-концевой стороны последовательности NLS крупного Т-антигена SV40, а линкерная последовательность GS присутствует между последовательностью NLS крупного Т-антигена SV40 и последовательностью NLS нуклеоплазмина.
[00343] В некоторых вариантах осуществления изобретения, белок Cas12а содержит линкерную последовательность GGGGSGGGGS и последовательность NLS крупного Т-антигена SV40 у С-конца, где последовательность NLS крупного Т-антигена SV40 расположена с С-концевой стороны линкерной последовательности GGGGSGGGGS.
[00344] В некоторых вариантах осуществления изобретения, белок Cas12а содержит последовательность, включающую линкер и NLS у С-конца. В некоторых вариантах осуществления изобретения, эта последовательность, включающая линкер и NLS, содержит аминокислотную последовательность, выбранную из SEQ ID NO: 479-490, или состоит из этой последовательности. В некоторых вариантах осуществления изобретения, эта последовательность, включающая линкер и NLS, содержит аминокислотную последовательность или состоит из аминокислотной последовательности, которая по меньшей мере на 80%, по меньшей мере на 85%, по меньшей мере на 90%, по меньшей мере на 91%, по меньшей мере на 92%, по меньшей мере на 93%, по меньшей мере на 94%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, или по меньшей мере на 99% идентична аминокислотной последовательности, выбранной из SEQ ID NO: 479-490.
[00345] Родственные комплексы направляющая Cas12a chRDNA/нуклеопротеин также могут быть получены с применением методов, хорошо известных специалистам в данной области. Белковые компоненты Cas12 могут быть получены рекомбинантно, а затем направляющие молекулы Cas12 chRDNA и белки Cas12 могут быть объединены в комплексы с применением методов, известных специалистам. См., например, Пример 2, в котором описан неограничивающий пример способа сборки нуклеопротеиновых комплексов, содержащих направляющую молекулу/белок Cas12.
[00345] Кроме того, могут быть разработаны клеточные линии, конститутивно экспрессирующие белки Cas12, которые могут быть трансфицированы направляющими компонентами Cas12 chRDNA, и эти комплексы могут быть очищены из клеток с применением стандартных методов очистки, таких как, но не ограничивающихся ими, аффинная, ионообменная и эксклюзионная хроматография. См., например, Jinek et al. (Science, 2012, 337:816-821).
[00347] Согласно известным способам, белки Cas12 могут быть получены с использованием экспрессионных кластеров, кодирующих белок Cas12. Экспрессионные кластеры обычно содержат регуляторные последовательности, которые являются функциональными в клетках-хозяевах, в которые их вводят. Регуляторные последовательности участвуют в одном или в нескольких из следующих процессов: в регуляции транскрипции, в посттранскрипционной регуляции и в регуляции трансляции. Экспрессионные кластеры могут присутствовать в экспрессионных векторах и могут быть введены в клетки-хозяева широкого спектра, включая бактериальные клетки, дрожжевые клетки, клетки растений и клетки млекопитающих.
[00348] Белки Cas12 могут быть продуцированы в векторах, включая экспрессионные векторы, содержащие полинуклеотиды, кодирующие белки Cas12. Векторы, подходящие для получения белков Cas12, включают плазмиды, вирусы (включая фаг) и интегрируемые фрагменты нуклеиновых кислот (то есть, фрагменты, интегрируемые в геном хозяина посредством гомологичной рекомбинации). Вектор реплицируется и функционирует независимо от генома хозяина или может, в некоторых случаях, интегрироваться в сам геном. Подходящие реплицирующиеся векторы будут содержать репликон и регуляторные последовательности, происходящие от видов, совместимых с предполагаемой экспрессией клетки-хозяина. В некоторых вариантах осуществления изобретения, полинуклеотид, кодирующий белок Cas12, функционально связан с индуцируемым промотором, репрессируемым промотором или конститутивным промотором. Экспрессионные векторы могут также включать полинуклеотиды, кодирующие белковые метки (например, полигистидиновые метки, гемагглютининовые метки, флуоресцентные белковые метки, биолюминесцентные метки, метки ядерной локализации). Кодирующие последовательности для таких белковых меток могут быть слиты с кодирующей последовательностью, или они могут быть включены в экспрессионный кластер, например, в нацеливающий вектор.
[00349] Общие способы конструирования экспрессионных векторов известны специалистам в данной области. Экспрессионные векторы для большинства клеток-хозяев имеются в продаже. Существует несколько коммерческих программных продуктов, разработанных для облегчения выбора подходящих векторов и их конструирования, таких как векторы клеток насекомых для трансформации клеток насекомых и экспрессии генов в клетках насекомых, бактериальные плазмиды для трансформации бактерий и экспрессии генов в бактериальных клетках, дрожжевые плазмиды для трансформации клеток и экспрессии генов в дрожжах и других грибах, векторы млекопитающих для трансформацию клеток млекопитающих и экспрессии генов в клетках млекопитающих или у самих млекопитающих, вирусные векторы (включая ретровирусные, лентивирусные и аденовирусные векторы) для трансформации клеток, а также для экспрессии генов; и способы, позволяющие легко клонировать такие полинуклеотиды. SnapGene™ (GSL Biotech LLC, Chicago, II1; snapgene.com/resources/plasmid_files/your_time_is_valuable/), например, предоставляет обширный список векторов, отдельных векторных последовательностей и векторных карт, а также коммерческие источники для многих векторов. Большое количество векторов млекопитающих, подходящих для применения, имеются в продаже (например, у Life Technologies, Grand Island, NY; NeoBiolab, Cambridge, MA; Promega, Madison, WI; ATUM, Menlo Park, CA; Addgene, Cambridge, MA).
[00350] Векторы, полученные из вирусов млекопитающих, также могут быть использованы для экспрессии белковых компонентов Cas12 способами согласно изобретению в клетках млекопитающих. К ним относятся векторы, полученные из таких вирусов, как аденовирус, аденоассоциированный вирус, парвовирус, вирус герпеса, полиомавирус, цитомегаловирус, лентивирус, ретровирус, вирус осповакцины и вирус обезьян 40 (SV40). См., например, Kaufman et al. (Mol. Biotech., 2000, 16:151-160); and Cooray et al. (Methods Enzymol., 2012, 507:29-57). Регуляторные последовательности, функционально связанные с последовательностями, кодирующими белок Cas12, могут включать последовательности связывания с активатором, энхансеры, интроны, последовательности распознавания полиаденилирования, промоторы, последовательности связывания с репрессором, структуры стебель-петля, последовательности инициации трансляции, лидерной последовательности трансляции, последовательности терминации транскрипции, последовательности терминации трансляции, сайты связывания с праймером и т.п. Обычно используемые промоторы представляют собой конститутивные промоторы млекопитающих CMV, MND, EF1a, SV40, PGK1 (мыши или человека), Ubc, CAG, CAMKIIa и бета-Act, и другие промоторы, известные специалистам в данной области. См., например, Khan et al. (Advanced Pharmaceutical Bulletin, 2013, 3: 257-263). Кроме того, могут быть использованы промоторы РНК-полимеразы III млекопитающих, включая H1 и U6.
[00351] Для экспрессии генных продуктов были использованы многочисленные клеточные линии млекопитающих, включая HEK 293 (клетки эмбриональной почки человека) и CHO (клетки яичника китайского хомячка). Эти клеточные линии могут быть трансфицированы стандартными методами (например, с использованием фосфата кальция или полиэтиленимина (PEI), или посредством электропорации). Другие типичные клеточные линии млекопитающих включают, но не ограничиваются ими, HeLa, U2OS, 549, HT1080, CAD, P19, NIH 3T3, L929, N2a, клетки эмбриональной почки 293 человека, MCF-7, Y79, SO-Rb50, Hep G2, DUKX-X11, J558L и клетки почек детеныша хомячка (BHK). Такие клетки являются примерами клеток, которые могут быть использованы для получения белков Cas12.
[00352] Векторы могут быть введены в прокариоту и могут распространяться в ней. Прокариотические векторы хорошо известны специалистам в данной области. Обычно, прокариотический вектор содержит ориджин репликации, подходящий для клетки-хозяина-мишени (например, oriC, полученный из E. Coli; pUC, полученный из pBR322; pSC101, полученный из сальмонеллы); источник 15A (полученный из p15A) и бактериальные искусственные хромосомы). Векторы могут включать селективный маркер (например, гены, кодирующие резистентность к ампициллину, хлорамфениколу, гентамицину и канамицину). Zeocin™ (Life Technologies, Grand Island, NY) может быть использован для отбора бактерий, грибов (включая дрожжи), растений и клеточных линий млекопитающих. Соответственно, могут быть сконструированы векторы, которые несут только один ген резистентности к лекарственному средству Zeocin™ в целях проведения отбора в ряде организмов. Специалистам известны подходящие промоторы для экспрессии белков у прокариот, например, T5, T7, рамнозы (индуцируемые), арабинозы (индуцируемые) и phoA (индуцируемые). Кроме того, промоторы T7 широко используются в векторах, которые также кодируют РНК-полимеразу T7. Прокариотические векторы могут также включать сайты связывания с рибосомами различной силы и сигналы секреции (например, mal, sec, tat, OmpC и pelB). Кроме того, векторы могут содержать промоторы РНК-полимеразы для экспрессии NATNA. Последовательности терминации транскрипции прокариотической РНК-полимеразы также хорошо известны (например, последовательности терминации транскрипции от S. pyogenes).
[00353] Экспрессия белков у прокариот часто осуществляется в E. coli с помощью векторов, содержащих конститутивные или индуцируемые промоторы, регулирующие экспрессию либо слитых, либо неслитых белков. Однако, экспрессия белка с использованием других прокариотических систем входит в объем настоящего изобретения.
[00354] В некоторых вариантах осуществления изобретения, вектор представляет собой вектор для экспрессии в дрожжах. Примеры векторов для экспрессии в Saccharomyces cerivisiae включают, но не ограничиваются ими: pYepSec1, pMFa, pJRY88, pYES2 и picZ. Способы экспрессии генов в дрожжевых клетках известны специалистам в данной области. См., например, Christine Guthrie and Gerald R. Fink («Guide to Yeast Genetics and Molecular and Cell Biology, Part A» in Methods in Enzymology, 2004, Volume 194, Elsevier Academic Press, San Diego, CA). Обычно, для экспрессии генов, кодирующих белок в дрожжах, требуется промотор, функционально связанный с представляющей интерес кодирующей областью, и терминатор транскрипции. Различные дрожжевые промоторы могут быть использованы для конструирования экспрессионных кластеров для экспрессии генов в дрожжах.
[00355] Геномное редактирование клеток с использованием комплексов направляющая Cas12a chRDNA/нуклеопротеин
[00355] Доставка направляющих молекул Cas12 CHRDNA, белков Cas12 и комплексов направляющая Cas12 chRDNA/нуклеопротеин согласно изобретению в клетки, in vitro, ex vivo или in vivo, может быть достигнута с применением ряда способов, известных специалисту в данной области. Неограничивающие способы введения этих компонентов в клетку включают доставку вирусного вектора, сонопорацию, сжатие клеток, электропорацию, нуклеофекцию, липофекцию, технологию распыления частиц с помощью «дробового ружья», бомбардировку микроструями или обработку химическими реагентами (например, проникающими в клетки пептидами).
[00357] В некоторых вариантах осуществления изобретения, электропорация может быть использована для доставки направляющих молекул Cas12 chRDNA согласно изобретению в клетки. Электропорация может быть также проведена для доставки комплексов направляющая Cas12 chRDNA/нуклеопротеин согласно изобретению. В этих способах, направляющие молекулы chRDNA или комплексы направляющая Cas12 chRDNA/нуклеопротеин смешивают в буфере для электропорации с клетками-мишенями с образованием суспензии. Затем эту суспензию подвергают воздействию электрического импульса с оптимизированным напряжением, который создает временные поры в фосфолипидном бислое клеточной мембраны, позволяя заряженным молекулам (таким как нуклеиновые кислоты и белки) проходить через поры и проникать в клетку. Реагенты и оборудование для проведения электропорации являются коммерчески доступными.
[00358] В Примере 3 проиллюстрирована нуклеофекция активированных Т-клеток комплексами направляющая Cas12 chRDNA/нуклеопротеин. В Примере 5 проиллюстрирована нуклеофекция активированных Т-клеток комплексами направляющая Cas12 chRDNA/нуклеопротеин.
[00359] Комплексы направляющая Cas12 chRDNA/нуклеопротеин могут быть использованы для расщепления нуклеиновой кислоты-мишени или связывания с ней. Направляющая молекула Cas12 chRDNA может быть введена в клетки с белком Cas12, что будет приводить к образованию комплекса направляющая Cas12 chRDNA/нуклеопротеин. Комплекс направляющая Cas12 chRDNA/нуклеопротеин может гибридизоваться с нуклеиновой кислотой-мишенью, где нуклеиновая кислота-мишень содержит PAM. В одном своем варианте, настоящее изобретение охватывает способ связывания последовательности нуклеиновой кислоты-мишени в полинуклеотиде (например, в двухцепочечной ДНК (дцДНК)), где указанный способ включает получение одного или более комплексов направляющая Cas12 chRDNA/нуклеопротеин для введения в клетку и доставку комплекса(ов) Cas12-нуклеопротеинов в клетку, что способствует облегчению контактирования комплекса(ов) направляющая Cas12 chRDNA/нуклеопротеин с последовательностью полинуклеотида-мишени. В одном варианте осуществления изобретения, первый комплекс направляющая Cas12 chRDNA/нуклеопротеин содержит направляющую молекулу Cas12 CHRDNA, имеющую первый элемент нацеливающей области, комплементарный первой последовательности-мишени нуклеиновой кислоты в полинуклеотиде; а второй комплекс направляющая Cas12 chRDNA/нуклеопротеин содержит направляющую молекулу Cas12 CHRDNA, имеющую вторую нацеливающая область, комплементарную второй последовательности-мишени нуклеиновой кислоты в полинуклеотиде. Контактирование комплексов направляющая Cas12 chRDNA/нуклеопротеин с полинуклеотидом приводит к связыванию комплексов направляющая Cas12 chRDNA/нуклеопротеин с последовательностями нуклеиновых кислот-мишеней в полинуклеотиде. В одном варианте осуществления изобретения, первый комплекс направляющая Cas12 chRDNA/нуклеопротеин связывается с первой последовательностью-мишенью нуклеиновой кислоты; а второй комплекс направляющая Cas12 chRDNA/нуклеопротеин связывается со второй последовательностью-мишенью нуклеиновой кислоты в полинуклеотиде.
[00360] Такие способы связывания последовательности-мишени нуклеиновой кислоты могут быть осуществлены in vitro (например, в биохимической реакции или в культивируемых клетках; в некоторых вариантах осуществления изобретения, культивируемые клетки представляют собой культивируемые клетки человека, которые остаются в культуре и не вводятся человеку); in vivo (например, в клетках живого организма, при условии, что в некоторых вариантах осуществления изобретения, таким организмом является не-человеческий организм); или ex vivo (например, в клетках, взятых у индивидуума, при условии, что в некоторых вариантах осуществления изобретения, индивидуум не является человеком).
[00361] Доставка направляющаих молекул Cas12 chRDNA, белков Cas12 и комплексов направляющая Cas12 chRDNA/нуклеопротеин согласно изобретению может быть достигнута путем упаковки компонентов в биологический компартмент. Биологический компартмент, содержащий эти компоненты, может быть введен in vivo (например, в клетках живого организма, при условии, что в некоторых вариантах осуществления изобретения, таким организмом не является человеческий организм). Биологические компартменты могут включать, но не ограничиваются ими, вирусы (лентивирус, аденовирус), наносферы, липосомы, квантовые точки, наночастицы, микрочастицы, нанокапсулы, везикулы, частицы полиэтиленгликоля, гидрогели и мицеллы.
[00362] Так, например, биологический компартмент может содержать липосому. Липосома может представлять собой самособирающуюся структуру, содержащую один или более липидных бислоев, каждый из которых может содержать два монослоя, включающих противоположно ориентированные амфипатические липидные молекулы. Амфипатические липиды могут содержать полярную (гидрофильную) головную группу, ковалентно связанную с одной или двумя или более неполярными (гидрофобными) ацильными или алкильными цепями. Энергетически несоответствующие контакты между гидрофобными ацильными цепями и окружающей водной средой индуцируют самосборку амфипатических липидных молекул, в результате чего полярные головные группы могут ориентироваться по направлению к поверхности бислоя, а ацильные цепи могут ориентироваться по направлению к внутренней части бислоя, что позволяет эффективно защищать ацильные цепи от их контактов с водной средой.
[00363] Примерами предпочтительных амфипатических соединений, используемых в липосомах, могут быть фосфоглицериды и сфинголипиды, репрезентативными примерами которых являются фосфатидилхолин, фосфатидилэтаноламин, фосфатидилсерин, фосфатидилинозит, фосфатидиновая кислота, фосфатидилглицерин, пальмитоилолеолилфосфатидилхолин, лизофосфатидилхолин, лизофосфатидилэтаноламин, димиристоилфосфатидилхолин (DMPC), дипальмитоилфосфатидилхолин (DPPC), диолеоилфосфатидилхолин, дистеароилфосфатидилхолин (DSPC), дилинолеоилфосфатидилхолин, яичный сфингомиелин или любые их комбинации.
[00364] Биологический компартмент может содержать наночастицу. Наночастица может иметь диаметр приблизительно от 40 нанометров до приблизительно 1,5 микрометров, приблизительно от 50 нанометров до приблизительно 1,2 микрометров, приблизительно от 60 нанометров до приблизительно 1 микрометра, приблизительно от 70 нанометров до приблизительно 800 нанометров, приблизительно от 80 нанометров до приблизительно 600 нанометров, приблизительно от 90 нанометров до приблизительно 400 нанометров, или приблизительно от 100 нанометров до приблизительно 200 нанометров. В некоторых случаях, по мере увеличения размера наночастиц, скорость высвобождения может быть замедленной или пролонгированной, а по мере уменьшения размера наночастиц, скорость высвобождения может быть повышенной.
[00365] В некоторых вариантах осуществления изобретения, комплексы направляющая Cas12 chRDNA/нуклеопротеин упакованы в биологический компартмент. В некоторых случаях, нуклеиновая кислота, кодирующая Cas12, и химически синтезированная направляющая chRDNA упакованы в биологический компартмент. В некоторых случаях, мРНК, кодирующая Cas12, и химически синтезированная направляющая chRDNA упакованы в биологический компартмент.
[00365] Специалистам в данноц области известно множество способов оценки и/или количественного определения взаимодействий между последовательностями нуклеиновых кислот и полипептидами, включая, но не ограничиваясь ими, следующие методы: анализы на иммунопреципитацию (ChIP), анализы на электрофоретический сдвиг подвижности ДНК (EMSA), анализы на высвобождение ДНК и анализы на захват и детектирование на микропланшетах. Коммерчески доступные наборы, материалы и реагенты являются подходящими для применения многих из этих методов и, например, могут быть приобретены у следующих поставщиков: Thermo Scientific (Wilmington, DE), Signosis (Santa Clara, CA), Bio-Rad (Hercules, CA) и Promega (Мadison, WI). Общим подходом к обнаружению взаимодействий между полипептидом и последовательностью нуклеиновой кислоты является EMSA (см., например, Hellman L. M., et al., Nature Protocols 2(8):1849-1861 (2007)).
[00367] В другом своем варианте, настоящее изобретение относится к способу разрезания последовательности-мишени нуклеиновой кислоты в полинуклеотиде (например, одноцепочечного разрезания в дцДНК или двухцепочечного разрезания в дцДНК), где указанный способ включает получение одного или более комплексов направляющая Cas12 chRDNA/нуклеопротеин для введения в клетку и доставку комплекса(ов) направляющая Cas12 chRDNA/нуклеопротеин в клетку в целях облегчения контактирования комплекса(ов) направляющая Cas12 chRDNA/нуклеопротеин с полинуклеотидом. В одном варианте осуществления изобретения, первый комплекс направляющая Cas12 chRDNA/нуклеопротеин содержит первую направляющую молекулу Cas12 CHRDNA, имеющую первую нацеливающую область, комплементарную первой последовательности-мишени нуклеиновой кислоты в полинуклеотиде; а второй комплекс направляющая Cas12 chRDNA/нуклеопротеин содержит вторую направляющую молекулу Cas12 CHRDNA, имеющую вторую нацеливающую область, комплементарную второй последовательности-мишени нуклеиновой кислоты в полинуклеотиде, и эти комплексы вводят в клетку. Контактирование приводит к разрезанию последовательности-мишени нуклеиновой кислоты в полинуклеотиде (например, дцДНК) посредством комплекса(ов) направляющая Cas12 chRDNA/нуклеопротеин. В одном варианте осуществления изобретения, первый комплекс комплекс направляющая Cas12 chRDNA/нуклеопротеин связывается с первой последовательностью-мишенью нуклеиновой кислоты в дцДНК и расщепляет первую цепь дцДНК; а второй комплекс направляющая Cas12 chRDNA/нуклеопротеин связывается со второй последовательностью-мишенью нуклеиновой кислоты в дцДНК и расщепляет вторую цепь дцДНК. В некоторых вариантах осуществления изобретения, последовательностью нуклеиновой кислоты-мишени является ДНК или геномная ДНК. Такие способы связывания последовательности нуклеиновой кислоты-мишени проводят in vitro, в клетке (например, в культивируемых клетках), ex vivo (например, в стволовых клетках, взятых у индивидуума) и in vivo.
[00368] Последовательность (последовательности) нуклеиновых кислот-мишеней может (могут) быть выбрана(ы) соответствующим образом на основе, например, желаемого местоположения в полинуклеотидной последовательности или в геноме; и/или желаемой последовательности гена в полинуклеотидной последовательности или в геноме, подлежащей удалению или дизрупции.
[00369] В дополнительном варианте разрезания последовательности-мишени нуклеиновой кислоты в полинуклеотиде, донорный полинуклеотид также может быть введен в клетку для облегчения включения по меньшей мере части донорного полинуклеотида в геномную ДНК клетки. Обычно, донорный полинуклеотид вводят в непосредственной близости от сайт-направленного разрыва нуклеиновой кислоты-мишени для улучшения инсерции (например, гомологичной рекомбинации) донорного полинуклеотида в сайт двухцепочечного разрыва. В некоторых случаях, донорный полинуклеотид вводят в непосредственной близости от сайта двухцепочечного разрыва в нуклеиновой кислоте-мишени посредством связывания его с белком Cas12, который генерирует двухцепочечный разрыв (например, Cas12a).
[00370] Донорная(ые) последовательность (последовательности) нуклеиновых кислот-мишеней может (могут) быть выбрана(ы) соответствующим образом на основе, например, желаемой модификации. Так, например, донорный полинуклеотид может кодировать весь представляющий интерес белок или его часть. В некоторых вариантах осуществления изобретения, донорный полинуклеотид может кодировать CAR.
[00371] Настоящее изобретение также охватывает доставку донорного полинуклеотида в клетку посредством вируса, где донорный полинуклеотид кодирует CAR.
[00372] В некоторых вариантах осуществления изобретения, донорный полинуклеотид может быть одноцепочечным. В некоторых вариантах осуществления изобретения, донорный полинуклеотид может быть двухцепочечным. В некоторых вариантах осуществления изобретения, донорная ДНК может представлять собой мини-кольцо. В некоторых вариантах осуществления изобретения, донорным полинуклеотидом может быть плазмида. В некоторых вариантах осуществления изобретения, плазмида может быть суперспирализованной. В некоторых вариантах осуществления изобретения, донорный полинуклеотид может быть метилированным. В некоторых вариантах осуществления изобретения, донорный полинуклеотид может быть неметилированным. Донорный полинуклеотид может содержать модификацию. Модификации могут включать модификации, описаннные в настоящей заявке, включая, но не ограничиваясь ими, биотинилирование, химическое конъюгирование и введение синтетических нуклеотидов.
[00373] Терапевтические композиции, применения и способы
[00374] Направляющие молекулы Cas12 chRDNA и комплексы направляющая Cas12 chRDNA/нуклеопротеин согласно изобретению могут быть использованы в целях получения модифицированных клеток (таких как CAR-экспрессирующие клетки). Такие модифицированные клетки могут быть использованы, например, в области клеточной терапии (например, для лечения или профилактики заболевания посредством введения клеток), а особенно для адоптивной клеточной терапии. Такими вводимыми клетками могут быть, например, генетически модифицированные адоптивные клетки. Генетические модификации могут быть введены в адоптивные клетки посредством направляющих молекул Cas12 chRDNA и комплексов направляющая Cas12 chRDNA/нуклеопротеин, описанных в настоящей заявке, с применением одного или нескольких способов доставки. Настоящее изобретение охватывает, например, модификацию и введение клеток, которые являются аутологичными или аллогенными для реципиента, которому они должны быть введены. Используемый здесь термин «аллогенный» относится к другому, генетически неидентичному индивидууму того же вида. Так, например, аллогенная клетка относится к клетке, полученной от другого, генетически неидентичного индивидуума того же вида (по отношению к реципиенту, которому вводят эту клетку). И напротив, термин «аутологичный» относится к одному и тому же индивидууму. Так, например, аутологичная клетка, вводимая индивидууму, относится к клетке (модифицированной или немодифицированной, или к ее модифицированному или немодифицированному потомству), которая происходит от того же индивидуума.
[00375] «Адоптивная клетка» означает клетку, которая может быть генетически модифицирована для применения в клеточной терапии. Адоптивные клетки включают, но не ограничиваются ими, стволовые клетки, индуцированные плюрипотентные стволовые клетки, эмбриональные стволовые клетки, стволовые клетки пуповинной крови, лимфоциты, природные клетки-киллеры, фибробласты, эндотелиальные клетки, эпителиальные клетки, клетки-предшественники поджелудочной железы и т.п.
[00375] «Стволовая клетка» относится к клетке, которая обладает способностью к самообновлению, то есть, способностью проходить многочисленные циклы клеточного деления при сохранении недифференцированного состояния. Стволовые клетки могут быть тотипотентными, плюрипотентными, мультипотентными, олигопотентными или унипотентными. Стволовые клетки представляют собой эмбриональные клетки, фетальные клетки, амниотические клетки, клетки взрослых индивидуумов или индуцированные плюрипотентные стволовые клетки.
[00377] «Индуцированная плюрипотентная стволовая клетка» (iPSC) относится к типу плюрипотентной стволовой клетки, которая была искусственно получена из неплюрипотентной клетки, обычно соматической клетки. В некоторых вариантах осуществления изобретения, соматическая клетка представляет собой соматическую клетку человека. Примеры соматических клеток включают, но не ограничиваются ими, фибробласты кожи, мезенхимальные клетки костного мозга, клетки сердечной мышцы, кератиноциты, клетки печени, клетки желудка, нервные стволовые клетки, клетки легких, клетки почек, клетки селезенки и клетки поджелудочной железы. Дополнительные примеры соматических клеток включают клетки иммунной системы, включая, но не ограничиваясь ими, В-клетки, дендритные клетки, гранулоциты, врожденные лимфоидные клетки, мегакариоциты, моноциты/макрофаги, миелоидные клетки-супрессоры, NK-клетки, Т-клетки, тимоциты и гемопоэтические стволовые клетки. Плюрипотентные стволовые клетки могут быть дифференцированы в клетки множества типов, включая соматические клетки, NK-клетки, NK-подобные клетки, Т-клетки, клетки, подобные Т-клеткам, NK-Т-клетки, клетки, подобные NK-Т-клеткам, дендритные клетки, клетки, подобные дендритным клеткам, макрофаги и клетки, подобные макрофагам. Плюрипотентные стволовые клетки могут быть отредактированы до или после дифференцировки с помощью комплекса направляющая Cas12 chRDNA/нуклеопротеин. iPSC могут быть дополнительно модифицированы до или после дифференцировки путем введения в геном экзогенного гена или последовательности, такой как последовательность, кодирующая CAR.
[00378] «Гемопоэтическая стволовая клетка» относится к недифференцированной клетке, которая обладает способностью дифференцироваться в гемопоэтическую клетку, такую как лимфоцит.
[00379] «Лимфоцит» относится к лейкоциту (белой кровяной клетке), который является частью иммунной системы позвоночных. Термин «лимфоцит» также охватывает гемопоэтическую стволовую клетку, которая дает начало лимфоидным клеткам. Лимфоциты включают Т-клетки для клеточно-опосредованного, цитотоксического адаптивного иммунитета, такие как цитотоксические CD4+- и/или CD8+-Т-клетки; альфа/бета-Т-клетки и гамма/дельта-Т-клетки; регуляторные Т-клетки, такие как Treg-клетки; природные клетки- киллеры (NK), которые участвуют в создании клеточно-опосредованного, цитотоксического врожденного иммунитета; и В-клетки для гуморального адаптивного иммунитета, регулируемого антителами; NK/T-клетки; клетки-киллеры, индуцируемые цитокинами (CIK-клетки); и антигенпрезентирующие клетки (APC), такие как дендритные клетки. Лимфоцит может представлять собой клетку млекопитающего, такую как клетка человека.
[00380] Под используемым здесь термином «лимфоцит» также подразумеваются Т-клетки, сконструированные на основе Т-клеточного рецептора (TCR), и эти клетки были генетически сконструированы для экспрессии одного или более специфических, встречающихся в природе или сконструированных Т-клеточных рецепторов, которые могут распознавать белковые или (глико)липидные антигены клеток-мишеней. Небольшие фрагменты этих антигенов, такие как пептиды или жирные кислоты, доставляются на поверхность клетки-мишени и презентируются Т-клеточным рецепторам как часть главного комплекса гистосовместимости (MHC). Связывание Т-клеточного рецептора с MHC, нагруженными антигеном, активирует лимфоцит.
[00391] Лимфоциты, инфильтрирующие опухоль (TIL), также входят в объем используемого здесь термина «лимфоцит». TIL представляют собой иммунные клетки, которые проникают в окружающую среду внутри и вокруг опухоли («в микроокружение опухоли»). TIL обычно выделяют из опухолевых клеток и микроокружения опухоли и отбирают in vitro по высокой реактивности против опухолевых антигенов. TIL культивируют in vitro в условиях, которые позволяют преодолеть толерантные воздействия, наблюдаемые in vivo, а затем вводят индивидууму для лечения.
[00382] Термин «лимфоцит» также охватывает генетически модифицированные Т-клетки и NK-клетки, такие как клетки, модифицированные для продуцирования химерных антигенных рецепторов (CAR) на поверхности Т- или NK-клеток (CAR-T-клетки и CAR-NK-клетки).
[00383] Лимфоциты могут быть выделены у индивидуума, такого как человек, например, из крови или из солидных опухолей, например, в случае TIL, или из лимфоидных органов, таких как тимус, костный мозг, лимфатические узлы и лимфоидные ткани, связанные со слизистой оболочкой. Способы выделения лимфоцитов хорошо известны специалистам в данной области. Так, например, лимфоциты могут быть выделены из мононуклеарных клеток периферической крови (МКПК), которые отделяют от цельной крови с использованием, например, фиколла, гидрофильного полисахарида, который разделяет слои крови, и центрифугирования в градиенте плотности. Обычно, образцы антикоагулянтной или дефибринизированной крови наносят поверх раствора Фиколла и центрифугируют для образования различных слоев клеток. Нижний слой включает красные кровяные клетки (эритроциты), которые собираются или агрегируются средой Фиколла и полностью оседают на дно. Следующий слой содержит в основном гранулоциты, которые также мигрируют вниз через раствор фиколла-пака. Следующий слой включает лимфоциты, которые обычно находятся на границе раздела между плазмой и раствором Фиколла, наряду с моноцитами и тромбоцитами. Чтобы выделить лимфоциты, этот слой извлекают, промывают солевым раствором для удаления тромбоцитов, фиколла и плазмы, а затем снова центрифугируют.
[00384] Другие методы выделения лимфоцитов включают биопэннинг, который позволяет выделять клеточные популяции из раствора посредством связывания представляющих интерес клеток с пластиковыми поверхностями, покрытыми антителами. Затем нежелательные клетки удаляют путем обработки специфическими антителами и комплементом. Кроме того, для обнаружения и подсчета лимфоцитов может быть проведен анализ с применением метода клеточного сортинга с активацией флуоресценции (FACS). В FACS-анализе используют проточный цитометр, который разделяет меченые клетки на основе различий в рассеянии света и флуоресценции.
[00385] В случае TIL, лимфоциты выделяют из опухоли и культивируют, например, в высоких дозах IL-2 и отбирают с помощью анализов методами совместного культивирования с высвобождением цитокинов, либо с использованием антител против аутологичной опухоли, либо против линий опухолевых клеток, соответствующих HLA. Культуры с признаками повышенной специфической реактивности по сравнению с аллогенным контролем, не соответствующим MHC, могут быть отобраны для быстрого размножения, а затем введены индивидууму для лечения рака. См., например, Rosenberg et al. (Clin. Cancer Res., 2011, 17:4550-4557); Dudly et al. (Science, 2002, 298:850-854); Dudly et al. (J. Clin. Oncol., 2008, 26:5233-5239); и Dudly et al. (J. Immunother., 2003, 26:332-342).
[00385] После выделения, лимфоциты могут быть охарактеризованы с точки зрения специфичности, частоты и функции. Часто используемые анализы включают анализ ELISPOT, который позволяет определить частоту Т-клеточного ответа.
[00387] После выделения, лимфоциты могут быть активированы с применением методов, хорошо известны специалистам в данной области, для стимуляции пролиферации и дифференцировки в специализированные эффекторные лимфоциты. Поверхностные маркеры для активированных Т-клеток включают, например, CD3, CD4, CD8, PD1, IL2R и т.п. Активированные цитотоксические лимфоциты могут уничтожать клетки-мишени после связывания родственных рецепторов на поверхности клеток-мишеней. Поверхностные маркеры для NK-клеток включают, например, CD16, CD56 и т.п.
[00388] После выделения и, возможно, активации, лимфоциты могут быть модифицированы с использованием комплекса направляющая Cas12 chRDNA/нуклеопротеин согласно изобретению в целях проведения адоптивной Т-клеточной иммунотерапии. В адоптивной иммунотерапии обычно используют иммунные клетки пациента (аутологичные клетки) для лечения рака. Однако, способы разработки адоптивной иммунотерапии согласно изобретению также допускают использование сторонних донорских клеток (аллогенных клеток), что позволяет разработать «готовые» методы лечения.
[00389] Таким образом, в некоторых вариантах осуществления изобретения, лимфоциты для использования в адоптивной иммунотерапии выделяют у индивидуума, модифицируют ex vivo, а затем повторно вводят тому же индивидууму. Этот метод известен как «аутологичная терапия лимфоцитами».
[00390] Альтернативно, лимфоциты могут быть выделены, модифицированы ex vivo и введены другому индивидууму. Этот метод известен как «аллогенная терапия лимфоцитами».
[00391] В некоторых вариантах осуществления изобретения, комплексы направляющая Cas12 chRDNA/нуклеопротеин используют для получения терапевтических композиций, содержащих аллогенные клетки. В предпочтительном варианте осуществления изобретения, аллогенные клетки представляют собой Т-клетки. В более предпочтительном варианте осуществления изобретения, Т-клетки экспрессируют CAR. В еще более предпочтительном варианте осуществления изобретения, CAR нацелен на антиген, связанный с раком.
[00392] В некоторых вариантах осуществления изобретения, Т-клетки могут быть модифицированы для обеспечения более безопасной и эффективной аллогенной терапии. Так, например, α-константа Т-клеточного рецептора (TRAC) представляет собой кодирующий белок ген, который образует часть αβ-TCR. Таким образом, выбранные мутации в TRAC, а также подавление экспрессии TRAC могут способствовать устранению реакции «трансплантат против хозяина» во время аллогенной клеточной терапии. См., например, Poirot et al. (Cancer Res., 2015, 75:3853-3864). Было показано, что доставка специфичного для CD19 CAR в локус TRAC с использованием системы CRISPR-Cas9 может привести к отторжению опухоли. См., например, Eyquem et al. (Nature, 2017, 543:113). Аналогично, β-константа Т-клеточного рецептора (TRBC) также может быть нацелена на предотвращение экспрессии αβ-TCR. См., например, Ren et al. (Clin. Cancer Res., 2017, 23:2255-2266).
[00393] Белок запрограммированной гибели клеток 1, также известный как PD1, PDCD1 и CD279, представляет собой рецептор клеточной поверхности, который играет важную роль в подавлении иммунной системы и способствует аутотолерантности посредством подавления воспалительной активности Т-клеток. PDCD1 связывается со своим родственным лигандом, «лигандом запрограммированной гибели 1», также известным как PD-L1, CD274 и гомолог 1 B7 (B7-H1). PD1 защищает от аутоиммунитета посредством двойного механизма стимулирования запрограммированной клеточной гибели (апоптоза) в антигенспецифичных Т-клетках в лимфатических узлах, с одновременным уменьшением апоптоза в противовоспалительных супрессорных Т-клетках (регуляторных Т-клетках). Посредством этих механизмов, связывание PD1 с PD-L1 ингибирует иммунную систему, предотвращая тем самым аутоиммунные расстройства, но при этом, не позволяет иммунной системе уничтожать раковые клетки. Соответственно, мутация или подавление продуцирования PD1 может оказаться полезной при Т-клеточной терапии.
[00392] PD1 является примером молекулы «иммунной контрольной точки». Молекулы иммунных контрольных точек служат для подавления или ингибирования иммунного ответа. Молекулы иммунной контрольной точки включают, но не ограничиваются ими, PD1, цитотоксический Т-лимфоцитарный антиген 4 (CTLA-4, также известный как CD152), LAG3 (также известный как CD223), Tim3 (также известный как HAVCR2), BTLA (также известный как CD272), BY55 (также известный как CD160), TIGIT (также известный как IVSTM3), LAIR1 (также известный как CD305), SIGLEC10, 2B4 (также известный как CD244), PPP2CA, PPP2CB, PTPN6, PTPN22, CD96, CRTAM, SIGLEC7, SIGLEC9, TNFRSF10B, TNFRSF10A, CASP8, CASP10, CASP3, CASP6, CASP7, FADD, FAS, TGFBRII, TGFRBRI, SMAD2, SMAD3, SMAD4, SMAD10, SKI, SKIL, TGIF1, IL10RA, IL10RB, HMOX2, IL6R, IL6ST, EIF2AK4, CSK, PAG1, SIT1, FOXP3, PRDM1, BATF, GUCY1A2, GUCY1A3, GUCY1B2 и GUCY1B3. В некоторых вариантах осуществления изобретения, одну или несколько молекул иммунной контрольной точки инактивируют с использованием комплексов направляющая Cas12 chRDNA/нуклеопротеин согласно изобретению. В некоторых вариантах осуществления изобретения, инактивацию одной или нескольких молекул иммунной контрольной точки объединяют с инактивацией одного или нескольких компонентов TCR, как описано выше.
[00395] Бета-2-микроглобулин (B2M) является компонентом молекул МНС класса I, присутствующих в ядро-содержащих клетках. Бета-2-микроглобулин выделяется клетками, включая опухолевые клетки, в кровь и необходим для сборки и экспрессии комплекса HLA I. Однако, экспрессия HLA на поверхности аллогенных Т-клеток вызывает быстрое отторжение Т-клетками иммунной системы хозяина. Таким образом, нарушение экспрессии бета-2-микроглобулина также желательно для повышения эффективности аллогенной Т-клеточной терапии. Кроме того, отсутствие экспрессии молекул MHC класса I на аллогенных Т-клетках вызывает клиренс иммунной системой хозяина. Таким образом, желательным является презентация только субпопуляции молекул HLA, а предпочтительно HLA-E, на поверхности клеток.
[00395] Дополнительные гены могут быть аналогичным образом нацелены с использованием описанных здесь направляющах молекул Cas12 chRDNA для повышения эффективности адоптивной иммунно-клеточной терапии. Неограничивающие примеры предпочтительных генов и хромосомных локализаций (сборка генома hg38) представлены в Таблице 4.
Мишени для адоптивной клеточной терапии
[00397] В некоторых вариантах осуществления изобретения, ген, кодирующий TRAC, нацеливают вовнутрь клетки с использованием направляющей Cas12 chRDNA, описанной в настоящей заявке. В некоторых вариантах осуществления изобретения, ген, кодирующий PD1, нацеливают вовнутрь клетки с использованием направляющей Cas12 chRDNA, описанной в настоящей заявке. В некоторых вариантах осуществления изобретения, ген, кодирующий B2M, нацеливают вовнутрь клетки с использованием направляющей Cas12 chRDNA, описанной в настоящей заявке. В некоторых вариантах осуществления изобретения, ген, кодирующий TRAC, и ген, кодирующий B2M, нацеливают вовнутрь клетки с использованием направляющих Cas12 chRDNA, описанных в настоящей заявке.
[00398] Клетки, модифицированные с использованием комплексов направляющая Cas12 chRDNA/нуклеопротеин согласно изобретению, могут быть использованы, например, в адоптивной клеточной терапии для лечения рака. В некоторых вариантах осуществления изобретения, модифицированная клетка представляет собой генетически модифицированный лимфоцит. Такие генетически модифицированные лимфоциты, такие как CAR-T-клетки, могут быть использованы для лечения различных типов рака у индивидуума, включая, но не ограничиваясь ими, рак предстательной железы; рак яичников; рак шейки матки; рак прямой и ободочной кишки; рак кишечника; рак яичек; рак кожи; рак легких; рак щитовидной железы; рак костей; рак молочной железы; рак мочевого пузыря; рак матки; рак влагалища; рак поджелудочной железы; рак печени; рак почек; рак головного мозга; рак спинного мозга; рак полости рта; опухоли околоушной железы; рак крови; лимфомы, такие как В-клеточные лимфомы; лейкозы и т.п. Предпочтительно, для такого лечения используют эффективное количество модифицированных клеток.
[00399] В Таблице 5 перечислены типичные В-клеточные лейкозы и лимфомы, поддающиеся лечению с использованием адоптивных клеток (таких как CAR-T-клетки), полученных с использованием комплексов направляющая Cas12 chRDNA/нуклеопротеин согласно изобретению. Следует отметить, что лимфоциты, модифицированные комплексами направляющая Cas12 chRDNA/нуклеопротеин, раскрытыми в настоящей заявке, используют для лечения заболеваний, неограничивающий список которых приводится ниже в Таблице 5.
В-клеточные лейкозы/лимфомы и их сокращения
[00400] В других вариантах осуществления изобретения, другие нарушения клеточной пролиферации можно лечить с использованием адоптивных клеток (таких как CAR-T-клетки), полученных с использованием комплексов направляющая Cas12 chRDNA/нуклеопротеин согласно изобретению, включая предраковые состояния; болезни крови; и иммунные расстройства, такие как аутоиммунные расстройства, включая, но не ограничиваясь ими, болезнь Аддисона, глютеновую болезнь, сахарный диабет типа 1, болезнь Грейвса, болезнь Хашимото, воспалительное заболевание кишечника, рассеянный склероз, псориаз, ревматоидный артрит, склеродермию и системную красную волчанку.
[00401] Процедуры адоптивной клеточной терапии, описанные в настоящей заявке, могут быть объединены в одно и то же или разное время с одной или несколькими дополнительными терапиями, выбранными из группы, состоящей из терапии антителами, химиотерапии, цитокиновой терапии, терапии дендритными клетками, генотерапии, гормональной терапии, лазерной терапии и лучевой терапии.
[00402] Введение модифицированных клеток согласно изобретению индивидуумам может быть осуществлено любым удобным способом, включая аэрозольную ингаляцию, инъекцию, прием вовнутрь, переливание, имплантацию или трансплантацию. Композиции, описанные в настоящей заявке, могут быть введены пациенту подкожно, интрадермально, вовнутрь опухоли, интранодально, интрамедуллярно, внутримышечно, путем внутривенной или внутрилимфатической инъекции или внутрибрюшинно.
[00403] В одном варианте осуществления изобретения, модифицированные клеточные композиции согласно изобретению предпочтительно вводят путем внутривенной инъекции. Введение может включать введение 104-109 клеток на кг массы тела, предпочтительно от 105 до 106 клеток/кг массы тела. Клетки могут быть введены в одной или нескольких дозах. В некоторых вариантах осуществления изобретения, эффективное количество модифицированных клеток вводят в виде однократной дозы. В других вариантах осуществления изобретения, эффективное количество клеток вводят в виде более чем одной дозы в течение определенного периода времени. Определение оптимальных диапазонов эффективных количеств клеток данного типа для лечения конкретного заболевания или состояния находится в пределах компетенции специалистов в данной области.
[00404] Клетки, содержащие химерный антигенный рецептор (CAR)
[00405] В некоторых вариантах осуществления изобретения, адоптивная клетка представляет собой CAR-экспрессирующую клетку. CAR представляет собой рецептор, сконструированный для распознавания специфического антигена или эпитопа и связывания с ними. Рецептор является химерным, поскольку он обладает как антигенсвязывающими, так и активирующими Т-клетки функциями. CAR обычно представляет собой слитый белок, содержащий внеклеточный лиганд-связывающий домен, способный связываться с антигеном, трансмембранный домен и по меньшей мере один внутриклеточный сигнальный домен. Внеклеточный лиганд-связывающий домен может содержать одноцепочечный вариабельный фрагмент (scFv), содержащий гибрид из двух или более вариабельных областей, соединенных одним или более линкерами. CAR может дополнительно содержать шарнирную область. CAR иногда называют «химерным рецептором», «Т-телом» или «химерным иммунным рецептором (CIR)».
[00405] В некоторых вариантах осуществления изобретения, CAR может представлять собой ТRUCK, универсальный CAR, самоуправляемый CAR, ТаnCAR, армированный CAR, CAR с аутодеструкцией, условный CAR, маркированный CAR, ТеnCAR, двойной CAR или sCAR.
[00407] TRUCK (Т-клетки, перенацеленные для универсального уничтожения цитокинами) совместно экспрессируют химерный антигенный рецептор (CAR) и противоопухолевый цитокин. Экспрессия цитокинов может быть конститутивной или индуцированной активацией Т-клеток. Нацеливание по специфичности CAR и локализованное продуцирование провоспалительных цитокинов обеспечивает рекрутинг эндогенных иммунных клеток в участки опухоли и может усиливать противоопухолевый ответ.
[00408] Универсальные аллогенные CAR-T-клетки сконструированы так, чтобы они больше не экспрессировали эндогенный Т-клеточный рецептор (TCR) и/или молекулы главного комплекса гистосовместимости (MHC), что позволяет предотвращать реакцию «трансплантат против хозяина» (GVHD) или отторжение, соответственно.
[00409] Самоуправляемые CAR совместно экспрессируют CAR и рецептор хемокина, который связывается с лигандом опухоли, что приводит к усилению хоминга опухоли.
[00410] CAR-T-клетки, сконструированные так, чтобы они были резистентными к иммуносупрессии (армированные CAR), могут быть генетически модифицированы так, чтобы они больше не экспрессировали различные молекулы иммунной контрольной точки (например, цитотоксический Т-лимфоцитарно-ассоциированный антиген 4 (CTLA4) или белок запрограммированной гибели клеток 1 (PD1)), а также рецептор переключения иммунной контрольной точки, либо они могут быть введены вместе с моноклональным антителом, которое блокирует передачу сигналов иммунной контрольной точки.
[00411] CAR с аутодеструкцией может быть сконструирован с использованием РНК, доставляемой посредством электропорации для кодирования CAR. Альтернативно, индуцируемый апоптоз Т-клеток может быть достигнут на основе связывания ганцикловира с тимидинкиназой в геномодифицированных лимфоцитах или на основе совсем недавно описанной системы активации каспазы 9 человека низкомолекулярным димеризатором.
[00412] Условная CAR-T-клетка по своей природе является нечувствительной или «выключенной» до тех пор, пока добавление небольшой молекулы не будет завершать цикл реакции, что будет обеспечивать полную трансдукцию как сигнала 1, так и сигнала 2, и тем самым активировать CAR-T-клетку. Альтернативно, Т-клетки могут быть сконструированы для экспрессии специфичного к адаптеру рецептора, обладающего аффинностью к вводимым впоследствии вторым антителам, направленным на антиген-мишень.
[00413] Маркированные CAR-T-клетки экспрессируют CAR и опухолевый эпитоп, с которыми связывается агент, а именно, моноклональное антитело. При возникновении неудаляемых побочных эффектов, введение моноклонального антитела приводит к клиренсу CAR-T-клеток и ослабляет симптомы без каких-либо дополнительных внепухолевых эффектов.
[00414] Тандемная CAR (TanCAR)-Т-клетка экспрессирует один CAR, содержащий два связанных scFv, которые обладают различными аффинностями и связаны с одним или несколькими внутриклеточными костимулирующими доменами и сигнальным доменом CD3ζ. Для активации TanCAR-T-клеток требуется присутствие только одного антигена на клетках-мишенях, однако, присутствие обоих антигенов облегчает синергическую активацию. В некоторых вариантах осуществления изобретения, scFv ТаnCAR содержит вариабельную область тяжелой цепи (VH) и вариабельную область легкой цепи (VL), пару из двух вариабельных областей тяжелой цепи (VH) или пару из двух вариабельных областей легкой цепи (VL). В другом варианте осуществления изобретения, два scFv TanCAR могут иметь многоуровневую конфигурацию. В еще одном варианте осуществления изобретения, два scFv TanCAR могут располагаться последовательно или в петлевой конфигурации. В конкретных вариантах осуществления изобретения, по меньшей мере один из scFv тандемного CAR представляет собой scFv против CD20, а второй scFv выбран для нацеливания на специфический антиген на раковых клетках, такой как scFv против BCMA, scFv против CD19, scFv против CD30, scFv против CD22, scFv против CD70, scFv против ROR1 или scFv против легкой цепи каппа.
[00415] Двойная CAR-T-клетка экспрессирует два отдельных CAR с различными мишенями для связывания лигандов; один CAR включает только домен CD3ζ, а другой CAR включает только костимулирующий(е) домен (домены). Активация двойных CAR-T-клеток требует совместной экспрессии обеих мишеней на опухоли.
[00415] Безопасный CAR (sCAR) состоит из внеклеточного scFv, слитого с внутриклеточным ингибирующим доменом, при этом, sCAR-T-клетки, совместно экспрессирующие стандартный CAR, активируются только при связывании с клетками-мишенями, которые имеют стандартную CAR-мишень, но не имеют sCAR-мишени.
[00417] Внеклеточный (распознающий антиген) домен CAR предпочтительно представляет собой одноцепочечное антитело, а более предпочтительно, scFv. В одном варианте осуществления изобретения, антигенсвязывающий домен содержит scFv. Однако, любой подходящий фрагмент, который связывается с данной мишенью с высокой аффинностью, может быть использован в качестве области распознавания антигена. Внеклеточный домен CAR, способный связываться с антигеном, может представлять собой, например, любой олигопептид или полипептид, который может связываться с определенным антигеном.
[00418] В зависимости от желаемого антигена, используемого в качестве мишени, CAR согласно изобретению может быть сконструирован так, чтобы он включал соответствующую антигенсвязывающую часть, которая является специфичной к желаемому антигену-мишени. Так, например, если BCMA является желаемым антигеном, который должен служить в качестве мишени, то антитело или фрагмент антитела (например, scFv), нацеленный на BCMA, может быть использован в качестве антигенсвязывающего фрагмента для включения в CAR согласно изобретению.
[00419] Предпочтительные клеточные мишени и scFv/белки, связывающиеся с CAR, которые нацелены на эти мишени, представлены ниже в Таблице 6.
Список репрезентативных клеточных мишеней и scFv против CAR
[00420] В определенных вариантах осуществления изобретения, клеточную мишень, с которой связывается CAR, более предпочтительно выбирают из BCMA, CD19, CD20, CD22, CD47, CD79b, CD371, ROR-1, EphA2, MUC16, глипикана 3, PSCA и клаудина 18.2.
[00421] В еще более предпочтительном варианте осуществления изобретения, клеточной мишенью, с которой связывается CAR, является BCMA.
[00422] В еще более предпочтительном варианте осуществления изобретения, клеточной мишенью, с которой связывается CAR, является CD371.
[00423] Внутриклеточный домен CAR может представлять собой олигопептид или полипептид, который, как известно, функционирует как домен, который передает сигнал, вызывающий активацию или ингибирование биологического процесса в клетке. Внутриклеточный домен может содержать активирующий домен, содержащий весь внутриклеточный сигнальный домен Т-клеточного рецептора или его часть (TCR) и/или корецептора, при условии, что он передает сигнал эффекторной функции. Цитоплазматические сигнальные последовательности, которые регулируют первичную активацию комплекса TCR, который действует стимулирующим образом, могут содержать сигнальные мотивы, которые известны как мотивы активации иммунорецепторов на основе тирозина (ITAM). Примеры ITAM, содержащих цитоплазматические сигнальные последовательности, включают последовательности, происходящие от CD8, CD3ζ, CD3δ, CD3γ, CD3ε, CD32 (FcyRIIa), DAP10, DAP12, CD79a, CD79b, FcyRIγ, FcyRIIIγ, FceRIβ (FCERIB) и FceRIγ (FCERIG).
[00424] В предпочтительных вариантах осуществления изобретения, домен активации внутриклеточного сигнального домена происходит от CD3ζ.
[00425] Внутриклеточный сигнальный домен CAR согласно изобретению может быть сконструирован так, чтобы он содержал домен активации, такой как сигнальный домен CD3ζ, либо отдельно, либо в комбинации с любым(и) другим(и) желательным(и) цитоплазматическим(и) доменом (доменами), подходящим(и) для CAR согласно изобретению. Так, например, внутриклеточный сигнальный домен CAR может содержать домен активации, такой как участок цепи CD3ζ, в дополнение к костимулирующему домену. Костимулирующий домен относится к части CAR, содержащей внутриклеточный домен костимулирующей молекулы.
[00425] Костимулирующая молекула представляет собой молекулу, отличающуюся от рецептора антигена или его лигандов, которые требуются для эффективного ответа лимфоцитов на антиген. Примеры таких костимулирующих молекул, где все молекулы или их часть могут быть использованы в костимулирующем домене CAR согласно изобретению, включают CD27, CD28, 4-1BB (CD137), OX40, CD30, CD40, ICOS-1, GITR, антиген-1, связанный с функцией лимфоцитов (LFA-1), CD2, CD7, LIGHT, NKG2C и B7-H3.
[00427] В предпочтительных вариантах осуществления изобретения, CAR содержит костимулирующий домен, происходящий по меньшей мере от 4-1BB.
[00428] Трансмембранный домен может быть получен как из природного, так и из синтетического источника. Если источник является природным, то домен может быть получен из любого мембраносвязанного или трансмембранного белка. Так, например, трансмембранная область может происходить от (то есть содержать по меньшей мере часть) трансмембранной(ых) области(областей) альфа-, бета- или дзета-цепи Т-клеточного рецептора, CD28, CD3ζ, CD3ε, CD45, CD4, CD5, CD8 (например, CD8α, CD8β), CD9, CD16, CD22, CD33, CD37, CD64, CD80, CD86, CD134, CD137 или CD154, KIRDS2, OX40, CD2, CD27, LFA-1 (CD11a, CD18), ICOS (CD278), 4-1BB (CD137), GITR, CD40, BAFFR, HVEM (LIGHTR), SLAMF7, NKp80 (KLRF1), CD160, CD19, IL2Rβ, IL2Rγ, IL7Rα, ITGA1, VLA1, CD49a, ITGA4, IA4, CD49D, ITGA6, VLA-6, CD49f, ITGAD, CD11d, ITGAE, CD103, ITGAL, CD11a, LFA-1, ITGAM, CD11b, ITGAX, CD11c, ITGB1, CD29, ITGB2, CD18, LFA-1, ITGB7, TNFR2, DNAM1 (CD226), SLAMF4 (CD244, 2B4), CD84, CD96 (тактильного), CEACAM1, CRTAM, Ly9 (CD229), CD160 (BY55), PSGL1, CD100 (SEMA4D), SLAMF6 (NTB-A, Ly108), SLAM (SLAMF1, CD150, IPO-3), BLAME (SLAMF8), SELPLG (CD162), LTBR и PAG/Cbp.
[00429] Альтернативно, трансмембранный домен может быть синтетическим, и в этом случае, он будет содержать преимущественно гидрофобные остатки, такие как лейцин и валин. В некоторых случаях, триплет фенилаланина, триптофана и валина будет обнаружен на каждом конце синтетического трансмембранного домена. Короткий олиго- или полипептидный линкер, например, длиной от 2 до 10 аминокислот, может образовывать связь между трансмембранным доменом и эндоплазматическим доменом CAR.
[00430] В предпочтительном варианте осуществления изобретения, трансмембранный домен происходит от CD8.
[00431] В некоторых вариантах осуществления изобретения, CAR имеет более, чем один трансмембранный домен, который может быть повтором одного и того же трансмембранного домена или может отличаться от других трансмембранных доменов.
[00432] Шарнирная область может содержать полипептидную шарнирную область различной длины, такую как область из одной или нескольких аминокислот, часть CD8 или часть IgG4, и их комбинации.
[00433] В предпочтительном варианте осуществления изобретения, шарнирная область происходит от CD8.
[00434] CAR также могут быть включены в TIL, NK-клетки, макрофаги, дендритные клетки, индуцированные плюрипотентные стволовые клетки (iPSC) или TCR, в результате чего образуются CAR-TIL, CAR-NK-клетки, CAR-M, CAR-DC и TCR-сконструированные CAR-T-клетки, соответственно. Описание CAR-T-клеток, способов получения CAR-T-клеток и их применения можно найти, например, у Brudno et al. (Nature Rev. Clin. Oncol., 2018, 15:31-46); Maude et al. (N. Engl. J. Med., 2014, 371:1507-1517); и Sadelain et al. (Cancer Disc., 2013, 3:388-398).
[00435] В некоторых вариантах осуществления изобретения, кластер для экспрессии CAR трансдуцируют в адоптивную клетку, и этот кластер интегрируется в сайт разрыва, опосредуемый белком Cas12. В Примере 9 настоящей заявки проиллюстрирована трансдукция первичных клеток вектором аденоассоциированного вируса (AAV), содержащим кластер CAR.
[00435] В некоторых вариантах осуществления изобретения, кластер для экспрессии CAR содержит промотор для стимуляции экспрессии CAR. Обычно используемые промоторы включают конститутивные промоторы млекопитающих CMV, MND, EF1a, SV40, PGK1 (мыши или человека), Ubc, CAG, CAMKIIa и бета-Act, и другие промоторы, известные специалистам в данной области. См., например, Khan et al. (Advanced Pharmaceutical Bulletin, 2013, 3:257-263). Альтернативно, кластер для экспрессии CAR может содержать последовательность вырезания рибосомы (также называемую самоотщепляющимся пептидом) и может быть введен с сохранением рамки считывания эндогенно экспрессируемого гена. Обычно используемые последовательности вырезания рибосомы включают T2A, P2A, E2A и F2A. Описание последовательностей вырезания рибосомы и их применения, можно найти, например, у Chng et al. (MAbs, 2015, 7(2):403-412). Аналогично, экспрессионные кластеры, не связанные с CAR, могут содержать аналогичные промоторы или последовательности вырезания рибосом.
[00437] В некоторых вариантах осуществления изобретения, комплексы направляющая Cas12a chRDNA/нуклеопротеин используют для лечения генетических заболеваний, вызываемых патогенными аутосомными «доминантно-негативными» мутациями, которые присутствуют на одном аллеле у пациента. В некоторых случаях, основной генетической мутацией может быть полиморфизм по одному нуклеотиду (SNP) на одном из аллелей. Комплексы направляющая Cas12a chRDNA/нуклеопротеин могут быть сконструированы для нацеливания на аллель SNP, но не на аллель дикого типа, что приводит к разрушению только аллеля SNP. См., например, Пример 7, в котором описан неограничивающий пример способа конструирования комплексов направляющая Cas12a chRDNA/нуклеопротеин для нацеливания на последовательность дикого типа, которые способны к снижению редактирования нецелевых последовательностей, содержащих SNP.
[00438] В некоторых вариантах осуществления изобретения, комплексы направляющая Cas12a chRDNA/нуклеопротеин могут быть использованы для селективного редактирования (например, нокаута или возврата к дикому типу с репарацией на основе гомологии) SNP-содержащего аллеля без модификации аллеля дикого типа. В некоторых вариантах осуществления изобретения, такое редактирование может приводить к дизрупции гена. В других вариантах осуществления изобретения, такое редактирование может восстанавливать аллель обратно в состояние «дикого типа», например, посредством репарации на основе гомологии. Так, например, ряд генетических заболеваний, которые приводят к прогрессирующей потере зрения, обусловлены патогенными аутосомными «доминантно-негативными» мутациями. Примеры стратегий коррекции SNP доминантно-негативного заболевания включают, но не ограничиваются ими, нацеливание на мутации SNP в гене родопсина, вызывающие пигментный ретинит, см., например, Li et al. (CRISPR J., 2018, 1(1):55-64); нацеливание на мутации SNP в трансформирующем факторе роста, кодируемого бета-индуцируемым (TGFBI) геном, вызывающие дистрофию роговицы, см., например, Christie et al. (Scientific Reports, 2017, 7(1):16174).
[00439] Комплексы направляющая Cas12a chRDNA/нуклеопротеин согласно изобретению могут быть доставлены, например, в ткани глаза, которые поражены аутосомными, патогенными «доминантно-негативными» генетическими мутациями. В некоторых вариантах осуществления изобретения, комплекс направляющая Cas12a chRDNA/нуклеопротеин предназначен для избирательной дизрупции аллеля заболевания, но при этом, без нацеливания на аллель дикого типа, в целях лечения основной патологии. Такие заболевания могут включать, но не ограничиваются ими, дистрофии желтого пятна, палочко-колбочковые дистрофии, колбочко-палочковидные дистрофии или хориоретинопатии. Очевидно, что раскрытые здесь комплексы направляющая Cas12a chRDNA/нуклеопротеин предназначены для лечения заболеваний, которыми являются, но не ограничиваются ими, генетические заболевания, вызывающие прогрессирующую потерю зрения.
[00440] Экспериментальная часть
Неограничивающие варианты осуществления изобретения проиллюстрированы в нижеследующих Примерах. Были предприняты попытки для обеспечения точности в отношении используемых чисел (например, количеств, концентраций, процентных изменений и т.п.), но следует учитывать некоторые экспериментальные ошибки и отклонения. Если это не оговорено особо, то температура указана в градусах по Цельсию, а давление равно или близко к атмосферному. Следует отметить, что эти Примеры приводятся только в качестве иллюстрации и не ограничивают объема различных вариантов осуществления изобретения. Не все из следующих стадий, изложенных в каждом Примере, являются обязательными, и порядок выполнения стадий в каждом Примере необязательно должен быть таким, как он представлен в настоящем описании. Буква «r», если она стоит перед нуклеотидом, указывает на РНК; а все остальные нуклеотиды представляют собой ДНК (см., например, Таблицы 15 и 17), если это не оговорено особо. Фосфортиоатные связи представлены «*» между соседними основаниями.
Пример 1
Получение цитотоксических Т-клеток (CD4+ и CD8+) из МКПК и культуры первичных клеток
[00441] В этом Примере проиллюстрировано получение CD4+- и CD8+-Т-клеток из донорских мононуклеарных клеток периферической крови (МКПК).
[00442] CD4+- и CD8+-Т-клетки получали из донорских МКПК в основном описанным ниже способом. Т-клетки выделяли из мононуклеарных клеток периферической крови (МКПК) с использованием RoboSep-S (STEMCELL Technologies, Cambridge, MA)) и набора для выделения человеческих Т-клеток EasySep™ (STEMCELL Technologies, Cambridge, MA) и активировали в течение 3 дней в присутствии гранул, содержащих антитела против CD3/CD28 (Dynabeads™; Gibco 11132D) в полной среде ImmunoCult-XF (в среде для размножения Т-клеток ImmunoCult-XF ((STEMCELL Technologies, Cambridge, MA), и в присутствии иммунных клеток CTS SR (Gibco A2596102), и антибиотиков-противогрибковых средств (100X, Corning 30-004-Cl)) с добавлением рекомбинантного человеческого (rh) IL-2 (100 единиц/мл). Через 3 дня, гранулы удаляли с помощью магнитной сепарации, и клетки размножали в течение 1 дня в полной среде ImmunoCult-XF, дополненной IL-2 (100 единиц/мл).
Пример 2
Клонирование, экспрессия, продуцирование и сборка комплексов направляющая Cas12a chRDNA/нуклеопротеин
[00443] В этом Примере описан способ клонирования, экспрессии и очистки комплексов направляющая Cas12a chRDNA/нуклеопротеин, а также способы получения компонентов направляющего Cas12a.
[00444] A. Клонирование белка Cas12
[00445] Каталитически активная последовательность белка Cas12a Acidaminococcus spp. (штамм BV3L6) (SEQ ID NO:1) была оптимизирована по кодону для экспрессии в клетках E. coli. На С-конце были добавлены линкер глицин-серин и одна последовательность ядерной локализации (NLS) (SEQ ID NO: 4). Олигонуклеотидные последовательности, кодирующие белок Cas12a-NLS (в следующих Примерах называемые белками AsCas12a и Cas12a), были переданы коммерческим производителям для синтеза. Затем последовательности ДНК были клонированы в подходящие векторы для экспрессии бактерий с применением стандартных методов клонирования.
[00445] B. Экспрессия и очистка белка Cas12a
[00447] Белок AsCas12a экспрессировали в E.coli с использованием экспрессионного вектора и очищали с помощью аффинной хроматографии, ионообменной хроматографии и эксклюзионной хроматографии, по существу, как описано, например, Swarts et al. (Molecular Cell, 2017, 66:221-233).
[00448] C. Продуцирование компонентов направляющих Cas12a
[00449] Направляющие Cas12a были получены путем связывания нацеливающей области с конкретной областью активации направляющих Cas12a. Нацеливающая область или спейсер предпочтительно содержит 20-нуклеотидную последовательность связывания с мишенью. Последовательность связывания с мишенью была комплементарна последовательности-мишени, которая находилась ниже (в 3'-направлении) от 5'-TTTV или 5'-TTTN PAM. Репрезентативными последовательностями области активации направляющей молекулы Cas12a являются SEQ ID NO:6, SEQ ID NO:8 и SEQ ID NO:10 для видов Acidaminococcus spp., L. bacterium и F. novicida Cas12a, соответственно.
[00450] Направляющие последовательности Cas 12a (такие как cr-РНК и chRDNA) были переданы коммерческому производителю для синтеза.
[00451] Компоненты направляющей РНК (такие как cr-РНК) могут быть получены посредством транскрипции in vitro (например, с использованием набора для синтеза РНК с быстрым высоким выходом T7; New England Biolabs, Ipswich, MA) из двухцепочечных (дц) ДНК-матриц путем включения промотора T7 у 5'-конца последовательностей матричной дцДНК.
[00452] D. Сборка комплекса направляющая Cas12a/нуклеопротеин
[00453] Cas12a (AsCas12a) Acidaminococcus spp., помеченный на С-конце последовательностью ядерной локализации (NLS), рекомбинантно экспрессировали в E. coli и очищали хроматографическими методами. Нуклеопротеиновые комплексы образовывались при концентрации 80 пмоль белка Cas12a:240 пмоль направляющей молекулы, если это не оговорено особо. Перед сборкой с белком Cas12a, каждый из направляющих компонентов (например, cr-РНК или chRDNA) доводили до желаемой общей концентрации (240 пмоль) в конечном объеме 1 мкл, инкубировали в течение 2 минут при 95°C, удаляли из термоячейки и оставляли для уравновешивания до комнатной температуры. Белок Cas12a разводили до соответствующей концентрации в буфере для связывания (60 мМ Трис-ацетата, 150 мМ ацетата калия, 30 мМ ацетата магния, при рН 7,9) до конечного объема 1,5 мкл и смешивали с 1 мкл направляющих компонентов с последующим инкубированием при 37°C в течение 10 минут.
Пример 3
Нуклеофекция Т-клеток (CD4+ и CD8+) из МКПК под действием комплексов направляющая Cas12a/нуклеопротеин
[00454] В этом Примере описана нуклеофекция активированных Т-клеток с помощью комплекса направляющая Cas12a/нуклеопротеин.
[00455] Комплексы направляющая Cas12a/нуклеопротеин Примера 2 трансфицировали в первичные активированные Т-клетки (CD4+ и CD8+) (полученные как описано в Примере 1) с использованием 96-луночной челночной системы Nucleofector™ (Lonza, Allendale, NJ). Комплекс направляющая Cas12a/нуклеопротеин распределяли в конечном объеме 2,5 мкл в отдельные лунки 96-луночного планшета. Суспендированные Т-клетки осаждали центрифугированием в течение 10 минут при 200×g, промывали фосфатно-буферным раствором, не содержащим кальция и магния (PBS), и клеточный осадок ресуспендировали в 10 мл PBS, не содержащего кальция и магния. Клетки подсчитывали с помощью автоматизированного счетчика клеток Countess® II Automated Cell Counter (Life Technologies; Grand Island, NY).
[00455] 2,27 клеток переносили в коническую пробирку объемом 15 мл и клетки осаждали. PBS удаляли путем аспирации, и клетки ресуспендировали в растворе Nucleofector™ P4 или Р3 (Lonza, Allendale, NJ) до плотности 25-16 клеток/мл на образец. Затем 20 мкл клеточной суспензии добавляли в каждую лунку, содержащую 2,5 мкл комплексов направляющая Cas12a/нуклеопротеин, и весь объем из каждой лунки переносили в лунку 96-луночного планшета Nucleocuvette™ (Lonza, Allendale, NJ). Планшет загружали на 96-луночный челнок Nucleofector™ (Lonza, Allendale, NJ), и клетки подвергали нуклеофекции с использованием программы CA137 Nucleofector™ (Lonza, Allendale, NJ). После нуклеофекции, 77,5 мкл полной среды ImmunoCult-XF с добавлением IL-2 (100 единиц/мл) добавляли в каждую лунку, и весь объем суспензии трансфицированных клеток переносили в 96-луночный планшет для культивирования клеток, содержащий 100 мкл предварительно подогретой полной среды ImmunoCult-XF с добавлением IL-2 (100 единиц/мл). Планшет переносили в инкубатор для культивирования тканей и выдерживали при 37°C в 5% CO2 в течение 48 часов перед последующим анализом.
Пример 4
Сборка человеческих генов с использованием комплексов направляющая Cas12a/нуклеопротеин
[00457] В этом Примере описано конструирование и использование комплексов направляющая Cas12a/нуклеопротеин для нацеливания на гены, кодирующие константную область альфа человеческих Т-клеток (TRAC), бета-2-микроглобулин (B2M), белок запрограммированной гибели клеток 1 (PDCD1), индуцируемый цитокинами SH2-содержащий белок (CISH) и прото-онкоген Cb1 B (CBL-B) в Т-клетках человека.
[00458] A. Конструирование направляющих молекул cr-РНК AsCas12a
[00459] Все последовательности из 20 нуклеотидов, расположенные ниже (в 3'-направлении) от мотива 5'-TTTV PAM в кодирующих областях генов, кодирующих человеческие TRAC, B2M, PDCD1, CISH и CBL-B, были отобраны для нацеливания (SEQ ID No: 12-189). Критериями выбора мишени являются, но не ограничиваются ими, гомология с другими областями генома; процентное содержание G-C; температура плавления; и присутствие гомополимера внутри спейсера.
[00460] Идентифицированные последовательности из 20 нуклеотидов были присоединены ниже (в 3'-направлении) последовательности активирующей области AsCas12a (SEQ ID NO:6).
[00461] Последовательности были переданы коммерческим производителям для синтеза. Затем отдельные комплексы направляющая Cas12a/нуклеопротеин получали как описано в Примере 2, и трансфицировали в первичные Т-клетки как описано в Примере 3.
[00462] B. Определение эффективности редактирования генома
[00463] (1) Генерацию последовательности дцДНК-мишени для глубокого секвенирования гДНК выделяли из первичных Т-клеток, подвергнутых нуклеофекции, через 48 часов после трансфекции с использованием комплексов направляющая Cas12a/нуклеопротеин, и 50 мкл раствора для экстракции ДНК QuickExtract™ (Epicentre, Madison, WI)) на лунку с последующим инкубированием при 37°C в течение 10 минут, 65°C в течение 30 минут и 95°C в течение 3 минут для остановки реакции. Выделенную гДНК разводили 50 мкл стерильной воды и образцы хранили при -80°C.
[00464] С использованием выделенной гДНК была проведена первая ПЦР с использованием смеси Q5 Hot Start High-Fidelity 2X Master Mix (New England Biolabs, Ipswich, MA) при концентрации 1×, праймеры, предназначенные для амплификации области вокруг мишени Cas12a, использовали по 0,5 мкМ каждого, и 3,75 мкл гДНК использовали в конечном объеме 10 мкл. Амплификацию проводили с помощью начального цикла при 98°C в течение 1 минуты, 35 циклов по 10 секунд при 98°C и 20 секунд при 60°C, 30 секунд при 72°C; и конечного удлинения при 72°C в течение 2 минут. Реакционные смеси для ПЦР разводили в соотношении 1:100 в воде.
[00465] Для облегчения мультиплексного секвенирования каждого образца использовали уникальный набор индексных праймеров для ПЦР со штрих-кодированием. ПЦР со штрих-кодированием проводили с использованием реакционной смеси, включающей смесь Q5 Hot Start High-Fidelity 2X Master Mix (New England Biolabs, Ipswich, MA) в концентрации 1×, праймеров по 0,5 мкМ каждого и 1 мкл 1:100 разведенной первой ПЦР-смеси в конечном объеме 10 мкл. Реакционные смеси амплифицировали следующим образом: при 98°C в течение 1 минуты, а затем путем проведения 12 циклов по 10 секунд при 98°C, 20 секунд при 60°C и 30 секунд при 72°C, с конечной реакцией удлинения при 72°C в течение 2 минут.
[00465] (2) Очистка SPRIselect
[00467] Реакционные смеси для ПЦР объединяли и переносили в одну центрифужную пробирку для очистки ампликонов на основе гранул SPRIselect (Beckman Coulter, Pasadena, CA) для секвенирования.
[00468] К ампликону добавляли 0,9 кратного объема гранул SPRIselect, перемешивали и инкубировали при комнатной температуре в течение 10 минут. Микроцентрифужную пробирку помещали на подставку для магнитных пробирок до тех пор, пока раствор не очищался. Супернатант удаляли и отбрасывали, а остаточные гранулы промывали 1 объемом 85% этанола, и эти гранулы инкубировали при комнатной температуре в течение 30 секунд. После инкубирования, этанол отсасывали, и гранулы сушили на воздухе при комнатной температуре в течение 10 минут. Микроцентрифужную пробирку снимали с магнитной подставки и 0,25 кратного объема буфера Qiagen EB (Qiagen, Venlo, Netherlands) добавляли к гранулам, энергично перемешивали и выдерживали в течение 2 минут при комнатной температуре. Микроцентрифужную пробирку возвращали на магнитную подставку, инкубировали до тех пор, пока раствор не очистится, и супернатант, содержащий очищенные ампликоны, переливали в чистую микроцентрифужную пробирку. Очищенные ампликоны определяли количественно с помощью системы Nanodrop™ 2000 System (Thermo Scientific, Wilmington, DE) и анализировали качество библиотеки с помощью системы Fragment Analyzer™ (Advanced Analytical Technologies, Ames, IA) и набора реагентов дцДНК DNF-910 (Advanced Analytical Technologies, Ames, IA).
[00469] (3) Методы глубокого секвенирования
[00470] Объединенные ампликоны были нормализованы до концентрации 4 нМ, вычисленной на основе значений системы Nanodrop™ 2000 и среднего размера ампликонов. Библиотеку анализировали на секвенаторе MiSeq (Illumina, San Diego, CA) с набором реагентов MiSeq v2 (Illumina, San Diego, CA) за 300 циклов с двумя 151-цикловыми запусками для парных концов и двумя 8-цикловыми считываниями индекса.
[00471] (4) Анализ данных глубокого секвенирования
[00472] Идентичность продуктов в анализе данных секвенирования определяли на основе индексных последовательностей штрих-кодов, адаптированных к ампликонам в ПЦР со штрих-кодированием. Для обработки данных MiSeq использовали вычислительную компьютерную программу, которая позволяет выполнять, например, задачи, описанные ниже:
a. Риды были выровнены по геному человека (конструкции GRCh38/38) с использованием программы Bowtie (bowtie-bio.sourceforge.net/index.shtml);
b. Выровненные риды сравнивали с ожидаемой последовательностью геномного локуса дикого типа, и риды, не совпадающие с какой-либо частью локуса дикого типа, отбрасывали;
c. Риды, соответствующие последовательности дикого типа, подсчитывали;
d. Риды с indel (инсерцией или делецией оснований) были классифицированы по типу indel и подсчитаны; и
e. Общее число ридов indel делили на сумму ридов дикого типа и ридов indel для получения процента мутированных ридов.
[00473] Посредством идентификации последовательностей indel в областях, на которые нацелены комплексы направляющая Cas12a/нуклеопротеин, была определена конечная эффективность редактирования генома под действием комплексов направляющая Cas12a/нуклеопротеин. Результаты эксперимента по редактированию внутри клетки приводятся ниже в Таблице 7.
Процент indel, детектированных с использованием комплексов cr-РНК Cas12a/нуклеопротеин
редактирования
редактирования
StDev=Стандартное отклонение, n=3
[00474] Данные, представленные в Таблице 7, продемонстрировали, что комплексы Cas12a cr-РНК/нуклеопротеин способны редактировать множество генов в первичных человеческих Т-клетках. Другие гены, такие как гены, которые описаны здесь в другом разделе, могут быть нацелены аналогичным образом с использованием AsCas12a или других белков Cas12a (таких как белки L. bacterium или F. novicida).
Пример 5
Конструирование направляющих молекул Cas12a chRDNA с использованием ДНК в последовательности связывания с мишенью
[00475] В следующем Примере описано конструирование направляющих молекул AsCas12a chRDNA для включения оснований ДНК в последовательность связывания с мишенью.
[00475] A. Конструирование направляющей молекулы in silico Cas12a chRDNA
[00477] Последовательность связывания с мишенью из 20 нуклеотидов направляющей молекулы AsCas12a была выбрана для конструирования, и в каждое положение последовательности связывания с мишенью было введено отдельное основание ДНК. Расположение оснований ДНК в последовательности связывания с мишенью направляющих молекул AsCas12a chRDNA показано в Таблице 8 (основания ДНК показаны буквой «d», а основания РНК показаны буквой «R»; также показана контрольная cr-РНК).
Последовательности связывания с направляющей молекулой-мишенью AsCas12a chRDNA
[00478] Три последовательности-мишени в гене, кодирующем B2M человека (B2M-tgt12, B2M-tgt1, B2M-интрон-tgt12), последовательность-мишень в гене, кодирующем TRAC человека (TRAC-tgt12), и последовательность-мишень в гене, кодирующем ДНК-метилтрансферазу 1 человека (DNMT1) (DNMT1-tgt1), были выбраны для редактирования. Направляющая молекула Cas12a chRDNA для каждой мишени, содержащей последовательность связывания с мишенью, включающую одно основание ДНК в каждом положении (см. Таблицу 8), а также контрольную последовательность Cas12a cr-РНК, были переданы коммерческому производителю для синтеза.
[00479] B. Трансфекция и анализ клеток
[00480] Отдельные комплексы направляющая Cas12a/нуклеопротеин для скрининга получали, по существу, как описано в Примере 2. Нуклеопротеиновые комплексы трансфицировали в первичные Т-клетки, как описано в Примере 3, и конечную эффективность редактирования генома комплексов комплексов направляющая Cas12a/нуклеопротеин определяли, как описано в Примере 4. Результаты эксперимента по редактированию внутри клетки представлены в Таблице 9.
Уровни редактирования посредством комплексов направляющая Cas12a/нуклеопротеин
StDev=стандартное отклонение; n=3
[00481] Результаты редактирования, представленные в Таблице 9, продемонстрировали, что направляющие молекулы Cas12a chRDNA, содержащие ДНК в спейсере, способны осуществлять редактирование на скорости, сравнимой с cr-РНК, по множеству мишеней (ср. SEQ ID NO: 211 с SEQ ID NO:224; SEQ ID NO: 232 с SEQ ID NO:234 или SEQ ID NO: 274 с SEQ ID NO: 293). Скорости редактирования направляющих конструкций chRDNА, представленные в Таблице 9, были нормализованы по скоростям редактирования cr-РНК для каждой мишени. Средние нормализованные скорости редактирования представлены на фиг. 13, где положение в последовательности связывания с мишенью представлено на графике в зависимости от среднего нормализованного редактирования. Предпочтительные положения оснований ДНК (то есть, более, чем 70% среднего нормализованного редактирования) обозначены серой заливкой и включают положения 1, 8, 9, 10, 11, 12, 14, 15, 16, 17, 18, 19 и 20. Данные, представленные в настоящем примере, а также данные, представленные в Таблице 9 и на фиг. 13, могут быть использованы для того, чтобы определить, какие положения в последовательности связывания с мишенью направляющей молекулы Cas12a могут быть сконструированы в виде ДНК.
Пример 6
Направляющие молекулы Cas12a chRDNA с несколькими основаниями ДНК в последовательности связывания с мишенью
[00482] В этом примере описано конструирование и тестирование направляющих молекул Cas12a chRDNA с несколькими основаниями ДНК в последовательности связывания с мишенью.
[00483] A. Конструирование In silico направляющих Cas12a chRDNA
[00484] Последовательность трех мишеней из 20 нуклеотидов в гене, кодирующем B2M человека (B2M-tgt12, B2M-tgt1, B2M-интрон-tgt12), мишени в гене, кодирующем TRAC человека (TRAC-tgt12), и мишени в гене, кодирующем ДНК-метилтрансферазу 1 человека (DNMT1-tgt1), были выбраны для редактирования. Для каждой мишени, от 1 до 7 нуклеотидов ДНК были введены в последовательность связывания с мишенью для каждой направляющей молекулы AsCas12a. Критерии конструирования для введения положения оснований ДНК включают, но не ограничиваются ими, полученные ранее данные скрининга одного положения (см. Пример 5), предварительное согласование положений, толерантных к ДНК (см. фиг. 13), расстояние между отдельными основаниями ДНК в последовательности связывания с мишенью и известное местоположение ошибочных спариваний в последовательности, не являющейся мишенью. Направляющие конструкции CHRDNA Cas12a, а также контрольная последовательность cr-РНК были переданы коммерческому производителю для синтеза.
[00485] B. Трансфекция и анализ клеток
[00485] Отдельные комплексы направляющая Cas12a/нуклеопротеин для скрининга получали, по существу, как описано в Примере 2. Комплексы направляющая Cas12a/нуклеопротеин трансфицировали в первичные Т-клетки, как описано в Примере 3, и конечную эффективность редактирования генома посредством комплексов направляющая Cas12a/нуклеопротеин определяли, как описано в Примере 4. Результаты эксперимента по редактированию внутри клетки и положение оснований ДНК в последовательности связывания с мишенью для каждой направляющей молекулы Cas12a chRDNA представлены в Таблице 10.
StDev=стандартное отклонение; n=3
[00487] Результаты редактирования, представленные в Таблице 10, продемонстрировали, что направляющие молекулы Cas12a chRDNA, содержащие множество оснований ДНК в последовательности связывания с мишенью, способны осуществлять редактирование со скоростью, сравнимой с cr-РНК для множества мишеней (ср. SEQ ID NO: 316 с SEQ ID NO: 321; SEQ ID NO: 330 с SEQ ID NO: 335, или SEQ ID NO: 345 с SEQ ID NO: 347).
Пример 7
Снижение скорости редактирования молекулы, не являющейся мишенью, посредством направляющих Cas12a chRDNA
В этом Примере описана идентификация нецелевых молекул Cas12a с помощью анализа SITE-Seq® (Cameron, P., et al., (2017). Mapping the genomic landscape of CRISPR-Cas9 cleavage. Nature Methods, 14(6), 600-606. https://doi.org/10.1038/nmeth.4284) и снижение скорости редактирования нецелевых молекул посредством направляющей Cas12a chRDNA по сравнению с направляющей cr-РНК Cas12a.
[00488] A. Анализ SITE-Seq®
[00489] Первичные Т-клетки человека культивировали как описано в примере 1. После размножения клеток в конических пробирках объемом 50 мл, из первичных Т-клеток человека выделяли высокомолекулярную геномную ДНК (гДНК) с использованием набора ДНК для культивирования крови и клеток Maxi Kit (Qiagen, Hilden, Germany) в соответствии с протоколом производителя.
[00490] Мишень из 20 нуклеотидов в гене, кодирующем человеческий рибосомный белок L32 (RPL32) (RPL32-tgt1), и в гене, кодирующем DNMT1 (DNMT1-tgt1), отбирали для оценки с помощью анализа SITE-Seq® (Cameron, P., et al., (2017). Nature Methods, 14(6), 600-606). Каждый направляющий компонент Cas12a последовательно разводили до соответствующей концентрации нуклеопротеина, инкубировали при 95°C в течение 2 минут, а затем оставляли для медленного нагревания до комнатной температуры в течение 5 минут. Нуклеопротеиновые комплексы Cas12a для мишеней RPL32 (SEQ ID NO: 375) и DNMT1 (SEQ ID NO: 404) получали путем объединения инкубированных направляющих Cas12a с белком Cas12a в соотношении 3:1 в буфере для реакции расщепления (60 мМ Трис-ацетата, 150 мМ ацетата калия, 30 мм ацетата магния, при рН 7,9) и инкубировали при 37°C в течение 10 минут. Отдельное расщепление 10 мкг геномной ДНК (гДНК) происходило при 6 концентрациях комплекса направляющая Cas12a/нуклеопротеин (8 нМ, 16 нМ, 32 нМ, 48 нМ, 64 нМ, 96 нМ и 128 нМ) в общем объеме 50 мкл. Реакции негативного контроля (0 нМ) проводили параллельно и в них не включали никакого нуклеопротеинового комплекса. Все реакции расщепления, включая негативный контроль, были собраны в трех экземплярах в 96-луночном планшете. Реакции расщепления проводили при инкубировании при 37°C в течение 4 часов.
[00491] Получение библиотеки и секвенирование проводили в основном как описано Cameron et al., (Nature Meth., 2017, 14:600-606), за исключением стадии образования хвоста dA после расщепления постнуклеопротеинового комплекса. Дополнительная стадия ферментативной репарации по концам была необходима из-за расщепления нуклеопротеинового комплекса Cas12a, приводящего к образованию ступенчатого (5'-выступающего) конца. Добавленные объемы оставались прежними, а исходные компоненты набора для образования хвоста dA были заменены на смесь ферментов End Prep (3 мкл) и 10-кратный буфер для реакции репарации по концам (5 мкл) из модуля NEBNext® Ultra™ End Repair/dA-Tailing от New England BioLabs (NEB # E7442L). Образцы инкубировали при 20°C в течение 30 минут, а затем при 65°C в течение 30 минут, и стандартную процедуру проводили снова. Секвенирование NGS проводили с использованием платформы Illumina NextSeq (Illumina, San Diego, CA), и для каждого образца было получено ~3 миллиона ридов. Любые сайты, полученные путем проведения анализа SITE-Seq® (Cameron, P., et al., (2017) Nature Methods, 14(6), 600-606) без нецелевых мотивов, расположенных в пределах 1 нуклеотида от сайта разрезания, считались ложноположительными и их отбрасывали. Количество восстановленных сайтов-мишеней, вычисленное путем проведения эксперимента по анализу молекул, не являющихся мишенью SITE-Seq®, показано в Таблице 11.
[00492] Данные, представленные в Таблице 11, продемонстрировали, что анализ SITE-Seq® (Cameron, P., et al., (2017). Mapping the genomic landscape of CRISPR-Cas9 cleavage. Nature Methods, 14(6), 600-606) позволил идентифицировать биохимические нецелевые молекулы направляющей Cas12a для последующей оценки внутри клетки.
[00493] B. Внутриклеточная валидация сайтов, полученных с помощью анализа SITE-Seq® на молекулы, не являющиеся мишенями
[00494] Для измерения частоты встречаемости indel в сайтах SITE-Seq®, не являющихся мишенями и представленных в Таблице 11, были проведены анализы на глубокое секвенирование мишеней в субпопуляции сайтов, выделенных в образцах RPL32 и DNMT1 для самых низких концентраций (например, 8 нМ и 16 нМ) нуклеопротеинового комплекса Cas12a. Два нецелевых сайта из образца RPL32 (SEQ ID NO: 371 и SEQ ID NO: 372) и один нецелевой сайт в DNMT1 (SEQ ID NO: 374) были отобраны для оценки частоты внутриклеточного нецелевого редактирования с помощью cr-РНК. Праймеры с прямым и обратным ампликоном были разработаны для каждого нецелевого участка и заказаны у коммерческого производителя.
[00495] Первичные Т-клетки человека культивировали, как описано в Примере 1. Нуклеопротеиновые комплексы RPL32 (SEQ ID NO: 375) и DNMT1 (SEQ ID NO: 404) Cas12a были получены, по существу, как описано в Примере 2. Нуклеопротеиновые комплексы переносили в первичные Т-клетки, как описано в Примере 3, и полученную в результате эффективность редактирования генома комплексов направляющая Cas12a/нуклеопротеин определяли, как описано в Примере 4. Нетрансфицированный пул клеток использовали в качестве эталона дикого типа. Мутантные риды (% инсерций-делеций) определяли как любые запросы неэталонных вариантов в пределах 20 пар оснований (п.о.) от сайта разрезания. При этом, отбрасывали сайты, которые имели низкий охват секвенирования (<1000 ридов в объединенных образцах, обработанных нуклеопротеиновым комплексом Cas12a, или <200 ридов в эталонных образцах) или >2% вариантов запроса в эталонных образцах. Сайты считались нецелевыми для клеток, если они накапливали >0,1% мутантных ридов в объединенных образцах, обработанных нуклеопротеиновым комплексом Cas12a. Результаты целевого глубокого секвенирования восстановленных нецелевых сайтов, проводенного с помощью SITE-Seq® (Cameron, P., et al., (2017). Nature Methods, 14(6), 600-606), представлены в Таблице 12, где нуклеотиды с ошибочным спариванием подчеркнуты.
StDev=стандартное отклонение; n=3
[00495] Данные, представленные в Таблице 12, продемонстрировали, что анализ SITE-Seq® позволил идентифицировать нецелевые сайты, которые были отредактированы в Т-клетках посредством направляющих cr-РНК Cas12a.
[00497] C. In silico-конструирование направляющих chRDNA
[00498] Для редактирования была выбрана мишень из 20 нуклеотидов в гене, кодирующем человеческий RPL32 (RPL32-tgt1), и в гене, кодирующем DNMT1 (DNMT1-tgt1). Для каждой мишени, от 1 до 4 нуклеотидов ДНК были сконструированы в последовательности связывания с мишенью каждой направляющей AsCas12a chRDNA. Критерии конструирования для введения положения оснований ДНК включают, но не ограничиваются ими, полученные ранее данные скрининга одного положения (см. Пример 5), предварительное согласование положений, толерантных к ДНК (см. фиг. 13), расстояние между отдельными основаниями ДНК в последовательности связывания с мишенью и известное местоположение ошибочных спариваний в последовательности, не являющейся мишенью. Направляющие конструкции CHRDNA Cas12a, а также контрольная последовательность cr-РНК Cas12a были переданы коммерческому производителю для синтеза.
[00499] D. Трансфекция и анализ клеток
[00500] Отдельные комплексы направляющая Cas12a/нуклеопротеин получали, по существу, как описано в Примере 2. Нуклеопротеиновые комплексы трансфицировали в первичные Т-клетки как описано в Примере 3, и полученную в результате эффективность редактирования генома посредством комплексов направляющая Cas12a/нуклеопротеин определяли, как описано в Примере 4. Результаты экспериментов по редактированию, а именно, по включению и выключению мишени (см. Таблицу 12) в клетках и по оценке расположения оснований ДНК в последовательности связывания с мишенью для каждой направляющей молекулы Cas12a chRDNA показаны для мишеней RPL32 (Таблица 13) и DNMT1 (Таблица 14). Кроме того были также протестированы направляющие молекулы Cas12a chRDNA с неосновным дезоксирибозным сайтом, а местоположение неосновного сайта показано как dN.
Направляющая RPL32 cr-РНК и chRDNA для редактирования мишеней и не-мишеней
ДНК
StDev=стандартное отклонение; n=3
Направляющая DNMT1 cr-РНК и chRDNA для редактирования мишеней и не-мишеней
StDev=стандартное отклонение; n=3
[00501] Результаты редактирования в Таблице 12 и в Таблице 13 продемонстрировали, что направляющие молекулы Cas12a chRDNA способны к снижению редактирования сайтов, не являющихся мишенями (ср. результаты в Таблице 13 для нецелевого редактирования SEQ ID NO: 375 с SEQ ID NO: 391; SEQ ID NO: 375 с SEQ ID NO: 394; ср. результаты в Таблице 14 для нецелевого редактирования SEQ ID NO: 404 с SEQ ID NO: 408; или SEQ ID NO: 404 с SEQ ID NO: 412), даже в сайтах с одним ошибочным спариванием нуклеотидов (см., например, Таблицу 12, SEQ ID NO: 371 и SEQ ID NO: 372).
Пример 8
Направляющие молекулы Cas12a chRDNA с основаниями ДНК в активирующей области
[00502] В этом примере описано конструирование и тестирование направляющих молекул AsCas12a chRDNA с основаниями ДНК в активирующей области.
[00503] A. Конструирование In silico направляющих молекул chRDNA
[00504] Активирующая область направляющей молекулы AsCas12a из 20 нуклеотидов проводника была выбрана для конструирования, где в каждом положении в активирующей области использовали отдельное основание ДНК. Расположение оснований ДНК в активирующей области направляющей молекулы AsCas12a показано в Таблице 15.
Направляющие молекулы AsCas12a chRDNA с ДНК в активирующей области
ДНК
[00505] Мишень в гене, кодирующем DNMT1 человека (SEQ ID NO: 361), была выбрана для редактирования. Последовательность связывания с мишенью DNMT была присоединена ниже (то есть, в 3'-направлении) к последовательностям активирующей области, содержащим по одному основанию ДНК в каждом положении (см. Таблицу 15). Последовательности были переданы коммерческому производителю для синтеза.
[00505] B. Трансфекция и анализ клеток
[00507] Отдельные комплексы направляющая Cas12a/нуклеопротеин для скрининга получали, по существу, как описано в Примере 2. Нуклеопротеиновые комплексы трансфицировали в первичные Т-клетки как описано в Примере 3, и полученную в результате эффективность редактирования генома посредством комплексов направляющая Cas12a/нуклеопротеин определяли, как описано в Примере 4. Результаты эксперимента по редактированию внутри клетки приводятся в Таблице 16.
Редактирование DNMT1 с использованием направляющих Cas12 chRDNA с ДНК в активирующей области
StDev=стандартное отклонение; n=3
[00508] Результаты редактирования, представленные в Таблице 16, продемонстрировали, что молекулы Cas12a chRDNA, содержащие ДНК в активирующей области, способны осуществлять редактирование на скорости, сравнимой с cr-РНК, по множеству мишеней (ср. SEQ ID NO: 438 с SEQ ID NO: 445; SEQ ID NO: 438 с SEQ ID NO:450 или SEQ ID NO: 438 с SEQ ID NO: 457). Скорости редактирования конструкций chRDNА, представленные в Таблице 16, были нормализованы по скоростям редактирования cr-РНК. Средние нормализованные скорости редактирования представлены на фиг. 14, где положение в активирующей области представлено на графике в зависимости от среднего нормализованного редактирования. Предпочтительные положения оснований ДНК (то есть, более, чем 70% среднего нормализованного редактирования) обозначены серой заливкой и включают положения 1, 3, 5, 7, 9, 10, 12, 13, 14, 15, 16, 17, 18 и 19.
[00509] D. CHRDNA с множеством оснований ДНК в активирующей области
[00510] На основании результатов, представленных в Таблице 10, отдельные положения ДНК были объединены в конструкции активирующих областей, содержащие множество оснований ДНК. Конструкция и расположение оснований ДНК в активирующей области направляющей AsCas12a показаны в Таблице 17.
Направляющие молекулы Cas12a chRDNA со множеством оснований ДНК в активирующей области
[00511] Конструкции активирующих областей, представленные в Таблице 17, были объединены с последовательностью-мишенью из 20 нуклеотидов в гене, кодирующем человеческий TRAC, сконструированный с использованием нуклеотидов ДНК в последовательности связывания с мишенью (SEQ ID NO: 321). Последовательность связывания с мишенью TRAC была присоединена ниже (то есть, в 3'-направлении) к последовательностям активирующей области, представленным в Таблице 17, а конструкции chRDNA, а также контрольная последовательность cr-РНК были переданы коммерческому производителю для синтеза.
[00512] E. Трансфекция и анализ клеток
[00513] Отдельные комплексы направляющая Cas12a/нуклеопротеин для скрининга получали, по существу, как описано в Примере 2. Отдельные комплексы направляющая Cas12a/нуклеопротеин трансфицировали в первичные Т-клетки как описано в Примере 3, и полученную в результате эффективность редактирования генома посредством комплексов направляющая Cas12a/нуклеопротеин определяли, как описано в Примере 4. Результаты эксперимента по внутриклеточному редактированию и расположение оснований ДНК в активирующей области и последовательности связывания с мишенью каждой chRDNA представлены в Таблице 18.
Редактирование TRAC с использованием направляющих молекул Cas12a chRDNA с ДНК в активирующей области и в последовательности связывания с мишенью
StDev=стандартное отклонение; n=3
[00514] Результаты редактирования в Таблице 18 продемонстрировали, что направляющие молекулы Cas12a chRDNA с ДНК как в активирующей области, так и в последовательности связывания с мишенью способны к редактированию со скоростью, сравнимой со скоростью для cr-РНК (ср. SEQ ID NO: 466 с SEQ ID NO:467; SEQ ID NO: 466 с SEQ ID NO: 468, или SEQ ID NO: 466 с SEQ ID NO: 469).
Пример 9
Клонирование донорного кластера AAV, получение AAV и AAV- трансдукция первичных клеток
[00515] В этом Примере описано конструирование и клонирование донорного кластера ДНК в вектор AAV, получение AAV, доставка комплексов направляющая Cas12a chRDNA/нуклеопротеин в первичные клетки и трансдукция первичных клеток посредством AAV для сайт-специфической интеграции экспрессионного кластера CAR в первичные клетки.
[00515] AAV может быть сконструирован для доставки донорных полинуклеотидов ДНК в клетки млекопитающих. Если доставка AAV происходит в комбинации с событием геномного расщепления, а донорный полинуклеотид ДНК в AAV фланкирован плечами гомологии, то донорный полинуклеотид ДНК может быть легко встроен в сайт геномного разрезания с помощью HDR, как описано, например, у Eyquem et al. (Nature, 2017, 543:113-117).
[00517] A. Конструирование донорных кластеров AAV In silico и получение rAAV
[00518] Была описана конструкция рецепторов CAR. См., например, Kochenderfer et al. (J. Immunotherapy, 2009, 32:689-702). Конструкция CAR была создана так, чтобы она содержала N-концевой сигнал секреции (сигнальный пептид CD8a), фрагмент scFv, специфичный для BCMA, за которым следуют шарнирная и трансмембранная области CD8, эффекторная область 4-1BB, эффекторная область CD3ζ и С-концевая сигнальная последовательность полиаденилирования BGH. Последовательность промотора млекопитающего была введена перед полинуклеотидом CAR. Для сайт-специфического встраивания донорных полинуклеотидов ДНК в геном клетки-хозяина после сайт-специфического расщепления был выбран сайт-мишень в эндогенном локусе TRAC (SEQ ID NO: 23). Затем были идентифицированы плечи гомологии длиной 500 п.о. у 5'- и 3'-сайта разрезания. 5'- и 3'-плечи гомологии были присоединены к концу донорных полинуклеотидов ДНК, где донорные полинуклеотиды ДНК были ориентированы в обратной ориентации (то есть, от 3' до 5') относительно плеч гомологии. Полученный донорный полинуклеотид ДНК представлен в SEQ ID NO: 413.
[00519] Была описана конструция антигена гистосовместимости B2M и HLA класса I, альфа-цепи E (HLA-E). См., например, Gornalusse et al. (Nature Biotechnology, 2017, 35(8):765-772). Конструкция слияния была сконструирована так, чтобы она содержала N-концевой сигнал секреции B2M, за которым следуют пептидная последовательность, происходящая от HLA-G, первая линкерная последовательность, последовательность B2M, вторая линкерная последовательность, последовательность HLA-E и С-концевая сигнальная последовательность полиаденилирования BGH. Последовательность промотора EF1α млекопитающих была встроена выше полинуклеотида B2M-HLA-E. Для сайт-специфического встраивания донорного полинуклеотида ДНК в геном клетки-хозяина после сайт-специфического расщепления был выбран сайт-мишень в эндогенном локусе В2М (SEQ ID NO: 62). Затем были идентифицированы плечи гомологии длиной 500 п.о. у 5'- и 3'-сайта разрезания. 5'- и 3'-плечи гомологии были присоединены к концу донорных полинуклеотидов ДНК, где донорные полинуклеотиды ДНК были ориентированы в обратной ориентации (то есть, от 3' до 5') относительно плеч гомологии. Полученный донорный полинуклеотид ДНК представлен в SEQ ID NO: 414.
[00520] Олигонуклеотидные последовательности, кодирующие донорные полинуклеотиды ДНК, были переданы коммерческому производителю для синтеза в подходящую рекомбинантную плазмиду AAV (rAAV). Плазмида rAAV, содержащая SEQ ID NO: 413, и отдельная плазмида rAAV, содержащая SEQ ID NO:4 14, были переданы коммерческому производителю для упаковки в два отдельных вируса AAV6.
[00521] B. Трансдукция первичных Т-клеток вирусом rAAV
[00522] Первичные активированные Т-клетки получали из МКПК, как описано в Примере 1. Комплексы направляющая Cas12a chRDNA/нуклеопротеин, нацеленные на гены, кодирующие TRAC (SEQ ID NO: 415) и B2M (SEQ ID NO: 416), получали, как описано в Примере 2. Т-клетки трансфицировали комплексами направляющая Cas12a chRDNA/нуклеопротеин, нацеленными на TRAC (SEQ ID NO: 415), и через период времени от 1 минуты до 4 часов после нуклеофекции, клетки инфицировали вирусом AAV6, упакованным донорной последовательностью CAR (SEQ ID NO: 413) при MOI 1×106. Кроме того, Т-клетки трансфицировали комплексами направляющая Cas12a chRDNA/нуклеопротеин, нацеленными на В2М (SEQ ID NO: 416), и через период времени от 1 минуты до 4 часов после нуклеофекции, клетки инфицировали вирусом AAV6, упакованным донорной последовательностью B2M-HLA-E (SEQ ID NO: 414) при MOI 1×106. Т-клетки культивировали в полной среде ImmunoCult-XF (STEMCELLS Technologies, Cambridge, МA), дополненной IL-2 (100 единиц/мл) в течение 24 часов после трансдукции. На следующий день, трансдуцированные Т-клетки переносили в конические 50 мл- пробирки и центрифугировали при 300×g в течение приблизительно 7-10 минут для осаждения клеток. Супернатант отбрасывали, а осадок осторожно ресуспендировали, и Т-клетки объединяли в соответствующем объеме полной среды ImmunoCult-XF (STEMCELLS Technologies, Cambridge, МA) с добавлением IL-2 (100 единиц/мл).
[00523] Перечисленные Т-клетки ресуспендировали в дозе 1×106 клеток/мл в полной среде ImmunoCult-XF (STEMCELLS Technologies, Cambridge, МA) с добавлением IL-2 (100 единиц/мл), и помещали в столько суспензионных колб T-175, сколько потребуется (максимальный объем одной колбы составляет 250 мл).
[00524] C. Экспрессия CAR-T-клеток, содержащих антитела против BCMA, B2M-HLA-E
[00525] Характеризацию in vitro CAR-T-клеток, содержащих антитела против BCMA, B2M-HLA-E, а также контрольных клеток (с нокаутом по TRAC (KO) и с нокаутом по B2M), и Т-клеток дикого типа проводили через 7 дней после трансдукции.
[00525] CAR-T-клетки оценивали на экспрессию посредством проточной цитометрии, а именно, либо на экспрессию CAR против BCMA с использованием рекомбинантного белка BCMA, конъюгированного с фикоэритрином (PE); экспрессию TRAC с использованием специфического антитела против TCR a/b, конъюгированного с Alexa Fluor® 647 (ThermoFisher Scientific, Waltham, MA), либо на экспрессию B2M с использованием специфического антитела против B2M, конъюгированного с PE. Результаты анализа посредством проточной цитометрии представлены на фиг. 15A, где CAR-позитивные (фиг. 15A, 1501), TRAC-позитивные (фиг. 15A, 1502) и B2M-позитивные результаты (фиг. 15A, 1503) показаны для клеток, которые не подвергались обработке (Т-клеток дикого типа; фиг. 15A, 1504), клеток, которые были трансфицированы только обоими комплексами направляющая Cas12a chRDNA/нуклеопротеин (TRAC KO/B2M KO; фиг. 15A, 1505), и клеток, которые были трансфицированы обоими комплексами направляющая Cas12a chRDNA/нуклеопротеин и трансдуцированы обоими вирусами (с анализом на антитела против BCMA, и B2M-HLA-E для CAR-T-клеток; фиг. 15A, 1506). На оси Y представлен процент позитивных клеток, определенный с помощью FACS, на различные маркеры клеточной поверхности. Результаты также представлены в Таблице 19.
Анализ с помощью проточной цитометрии для Cas12a/AAV-обработанных клеток
[00527] Результаты, представленные на фиг. 15A и в Таблице 19, демонстрируют нокаут экспрессии эндогенной TRAC и B2M, опосредованный направляющей Cas12a chRDNA, и введение и экспрессию CAR против BCMA и экзогенных белков B2M-HLA-E, опосредуемые AAV6.
[00528] D. Цитотоксичность CAR-T клеток, содержащих антитела против BCMA, B2M-HLA-E in vitro
[00529] Цитотоксичность CAR-T клеток, содержащих антитела против BCMA, B2M-HLA-E, оценивали in vitro в отношении цитотоксичности линии клеток множественной миеломы NCI-H929, которые презентируют антиген BCMA. Т-клетки с нокаутом TRAC использовали в качестве контроля для CAR-опосредованного цитолиза. Вкратце, клетки-мишени (NCI-H929 (T)) метили фиолетовым CellTrace™ (CTV; Thermo Fisher C34557) для того, чтобы отличить эти клетки от эффекторных CAR-T-клеток, содержащих антитела против BCMA, B2M-HLA-E (E), и клетки совместно культивировали в соотношении E:T, равном 0:1, 1:20, 1:10, 1:5, 1:3, 1:1, 3:1 и 10:1 (3 лунки для совместного культивирования/соотношение E:T). Цитотоксичность измеряли путем стробирования популяции клеток CTV (клеток-мишеней) и живых клеток, оцененного с использованием иодида пропидия (PI) через 48 часов совместного культивирования. Данные анализировали методом проточной цитометрии (Intellicyt iQue Screener Plus). Специфический лизис вычисляли по следующему уравнению для каждой лунки: Специфический лизис=1 - (количество живых клеток-мишеней в тестируемом образце/количество живых клеток-мишеней в контрольном образце).
[00530] Результаты анализа на цитотоксичность in vitro представлены на фиг. 15B для CAR-T-клеток, содержащих антитела против BCMA, B2M-HLA-E (серые кружки) и для контрольных T-клеток с нокаутом по TRAC (черные кружки). Ось Y представляет процент уничтожения клеток-мишеней, а ось X указывает на используемое соотношение E:T. Данные, представленные на фиг. 15B, также представлены в Таблице 20.
Цитотоксичность, опосредуемая CAR против BCMA
StDev=стандартное отклонение; n=3
[00531] Результаты, представленные на фиг. 15B и в Таблице 20, продемонстрировали, что CAR-T-клетки, полученные с использованием направляющих молекул Cas12a chRDNA, способны к антигенспецифическому уничтожению клеток-мишеней.
[00532] Представленные здесь способы могут быть применены для получения других клеток с использованием направляющих молекул Cas12a chRDNA для сайт-специфического введения донорных полинуклеотидов, содержащих химерный антигенный рецептор (CAR). Дополнительные донорные полинуклеотиды, экспрессирующие полипептиды, не являющиеся CAR (то есть, конструкции слияния B2M-HLA-E), могут быть аналогичным образом введены в соответствии с приведенным здесь руководством.
Пример 10
Получение CAR-T-клеток, содержащих антитела против BCMA и экспрессирующих слитую конструкцию В2M-HLA-E под контролем эндогенного промотора B2M
[00533] В этом примере описана конструкция для трансдукции первичных клеток вирусом AAV для сайт-специфической интеграции полинуклеотида CAR и экспрессионного кластера полинуклеотида B2M-HLA-E в сайтах разрыва, опосредованных Cas12a chRDNA, в геном первичных клеток.
[00534] A. Конструирование донорных кластеров AAV in silico и продуцирование rAAV
[00535] CAR против BCMA был конструировали как описано в Примере 9.
[00535] Донорный кластерный полинуклеотид для конструкции слияния P2A-B2M-HLA-E был сконструирован с использованием полинуклеотида, кодирующего N-концевой сигнал секреции B2M, за которым следуют: полинуклеотид, кодирующий пептидную последовательность, происходящую от HLA-G; полинуклеотид, кодирующий первую линкерную последовательность; полинуклеотид, кодирующий последовательность B2M; полинуклеотид, кодирующий вторую линкерную последовательность; полинуклеотид, кодирующий последовательность HLA-E; и полинуклеотид, кодирующий С-концевую сигнальную последовательность полиаденилирования BGH. Полинуклеотид, кодирующий последовательность вырезания рибосомы P2A, встраивали перед B2M-HLA-E так, чтобы экспрессия слитой конструкции находилась под контролем эндогенного промотора B2M. Для сайт-специфического встраивания донорных полинуклеотидов ДНК в геном клетки-хозяина после сайт-специфического расщепления был выбран сайт-мишень в эндогенном локусе B2M (SEQ ID NO: 62). Затем были идентифицированы плечи гомологии длиной 500 пар оснований у 5'- и 3'-концов сайта разрезания. 5'- и 3'-плечи гомологии были присоединены к 5'- и 3'-концам донорных полинуклеотидов ДНК, где донорные полинуклеотиды ДНК были ориентированы в прямом направлении (то есть, от 5' до 3') относительно плеч гомологии. Полученный донорный полиноклеотид ДНК представлен в SEQ ID NO: 479.
[00537] Олигонуклеотидные последовательности, кодирующие донорные полинуклеотиды ДНК, были переданы коммерческому производителю для синтеза в подходящую рекомбинантную плазмиду AAV (rAAV). Плазмида rAAV, содержащая SEQ ID NO:413, и отдельная плазмида rAAV, содержащая SEQ ID NO:479, были переданы коммерческому производителю для упаковки в два отдельных вируса AAV6.
[00538] B. Трансдукция первичная Т-клеток вирусом rAAV
[00539] Первичные активированные Т-клетки получали из МКПК, как описано в Примере 1. Комплексы направляющая Cas12a chRDNA/нуклеопротеин, нацеленные на гены, кодирующие TRAC (SEQ ID NO: 415) и B2M (SEQ ID NO: 416), получали, как описано в Примере 2. Т-клетки трансфицировали комплексами направляющая Cas12a chRDNA/нуклеопротеин, нацеленными на TRAC (SEQ ID NO: 415), и через период времени от 1 минуты до 4 часов после нуклеофекции, клетки инфицировали вирусом AAV6, упакованным донорной последовательностью CAR (SEQ ID NO: 413) при MOI 1×106. Кроме того, Т-клетки трансфицировали комплексами направляющая Cas12a chRDNA/нуклеопротеин, нацеленными на В2М (SEQ ID NO: 416), и через период времени от 1 минуты до 4 часов после нуклеофекции, клетки инфицировали вирусом AAV6, упакованным донорной последовательностью Р2А-B2M-HLA-E (SEQ ID NO: 479) при MOI 1×106. Т-клетки культивировали в полной среде ImmunoCult-XF (STEMCELLS Technologies, Cambridge, МA), дополненной IL-2 (100 единиц/мл) в течение 24 часов после трансдукции. На следующий день, трансдуцированные Т-клетки переносили в конические 50 мл- пробирки и центрифугировали при 300×g в течение приблизительно 7-10 минут для осаждения клеток. Супернатант отбрасывали, а осадок осторожно ресуспендировали, и Т-клетки объединяли в соответствующем объеме полной среды ImmunoCult-XF (STEMCELLS Technologies, Cambridge, МA) с добавлением IL-2 (100 единиц/мл).
[00540] Перечисленные Т-клетки ресуспендировали в дозе 1×106 клеток/мл в полной среде ImmunoCult-XF (STEMCELLS Technologies, Cambridge, МA) с добавлением IL-2 (100 единиц/мл), и помещали во столько суспензионных колб T-175, сколько потребуется (максимальный объем одной колбы составляет 250 мл).
[00541] C. Экспрессия CAR-T-клеток, содержащих антитела против BCMA, B2M-HLA-E
[00542] Характеризацию in vitro CAR-T-клеток, содержащих антитела против BCMA, Р2А-B2M-HLA-E, а также контрольных клеток (с нокаутом по TRAC (KO) и с нокаутом по B2M), и Т-клеток дикого типа проводили через 7 дней после трансдукции.
[00543] CAR-T-клетки оценивали на экспрессию посредством проточной цитометрии, а именно, либо на экспрессию CAR против BCMA с использованием рекомбинантного белка BCMA, конъюгированного с фикоэритрином (PE); экспрессию TRAC с использованием специфического антитела против TCR a/b, конъюгированного с Alexa Fluor® 647 (ThermoFisher Scientific, Waltham, MA), либо на экспрессию B2M с использованием специфического антитела против B2M, конъюгированного с PE. Результаты анализа посредством проточной цитометрии представлены в Таблице 21, где CAR-позитивные, TRAC-позитивные и B2M-позитивные результаты показаны для клеток, которые не подвергались обработке (Т-клеток дикого типа), клеток, которые были трансфицированы только обоими комплексами направляющая Cas12a chRDNA/нуклеопротеин (TRAC KO/B2M KO), и клеток, которые были трансфицированы обоими комплексами направляющая Cas12a chRDNA/нуклеопротеин и трансдуцированы обоими вирусами (с анализом на антитела против BCMA и B2M-HLA-E для CAR-T-клеток).
Анализ с помощью проточной цитометрии для Cas12a/AAV-обработанных клеток
[00544] Результаты, представленные в Таблице 21, продемонстрировали нокаут экспрессии эндогенной TRAC и B2M, опосредованный направляющей Cas12a chRDNA и введение и экзогенную экспрессию донорного кластера CAR против BCMA и экспрессию экзогенного донорного кластера B2M-HLA-E под контролем эндогенного промотора B2M.
[00545] Методы, представленные в настоящей заявке, могут быть применены для идентификации направляющих конструкций Cas12 chRDNA для других мишеней. Активирующая область и последовательности связывания с мишенью других направляющих Cas12 chRDNA могут быть подвергнуты скринингу методами, аналогичными описанным здесь методам.
Пример 11
Комплексы направляющая Cas12a/нуклеопротеин с альтернативной конфигурацией линкер-NLS
[00545] В этом примере описаны конструирование и сравнение комплексов направляющая Cas12a/нуклеопротеин, имеющих различные конфигурации линкера и сигнала ядерной локализации (NLS), с «неоптимизированной» конструкцией, содержащей глицин-сериновые линкеры и NLS крупного Т-антигена вируса вакуолизации обезьян 40 (SV40; SEQ ID NO:04), и используемой в предыдущих Примерах данной заявки.
[00547] A. Конструирование in silico последовательностей линкера Cas12a-NLS
[00548] Белок Cas12a (SEQ ID NO:01) Acidaminococcus sp. (штамм BV3L6) был выбран для конструирования, и две последовательности NLS, SV40 (SEQ ID NO:04) и последовательность нуклеоплазмина (NPL; SEQ ID NO:05) были выбраны для ковалентного присоединения к белку Cas12a с использованием аминокислотных линкеров глицин-серин (GS) или пар аминокислотных линкеров глицин-глицин-глицин-глицин-серин (G4S). Для тестирования также были созданы конструкции, включающие две NLS с вариабельными линкерами. Конструкции последовательностей линкер-NLS представлены в Таблице 22:
Конструкции последовательностей линкер-NLS
[00549] Последовательности NLS, представленные в Таблице 22, были клонированы на С-конце белка Cas12a (SEQ ID NO:01) Acidaminococcus sp. (штамм BV3L6), и рекомбинантный белок экспрессировали как описано в Примере 2.
[00550] B. Клеточная активность конструкций линкер-NLS Cas12a
[00551] Очищенный рекомбинантный белок Cas12a, содержащий последовательности линкер-NLS, представленные в Таблице 22, образовывал комплекс с направляющей chRDNA, нацелннной на ген TRAC (SEQ ID NO:467), как описано в Примере 2, и эти комплексы переносили в первичные Т-клетки как описано в Примере 3. Через 48 часов после трансфекции, конечную эффективность редактирования генома каждого комплекса направляющая Cas12a chRDNA/нуклеопротеин определяли как описано в Примере 4. Результаты эксперимента по редактированию клеток показаны на фиг. 16А и представлены в Таблице 23.
Скрининг на редактирование линкера-NLS Cas12a в клетках
StDev=стандартное отклонение; n=3; *=конструкция NLS, используемая в предыдущих примерах
[00552] Данные, представленные на фиг. 16A, а также в Таблице 23 настоящего примера, показали, что альтернативные линкеры и последовательности NLS могут обеспечивать усиление редактирования по сравнению с конструкцией, имеющей одну конфигурацию NLS GS-SV40.
[00553] C. Клеточная активность альтернативных последовательностей линкера Cas12a-NLS по отношению к мишеням
[00554] Четыре основные конфигурации линкера Cas12a-NLS, представленные в Таблице 23 (SEQ ID NO:489, SEQ ID NO:485, SEQ ID NO:487 и SEQ ID NO:483), и «неоптимизированную» конструкцию (SEQ ID NO:479) отбирали для сравнения с использованием смешанной панели cr-РНК и chRDNA. Нацеливающая область, включая положение оснований ДНК в конструкциях chRDNA, представлена в Таблице 24.
Мишени Cas12a для сравнения с линкером-NLS
[00555] Нацеливающие области, представленные в Таблице 24, были присоединены к 3'-концу активирующей области (SEQ ID NO:459) и переданы коммерческому производителю для синтеза.
[00555] Отдельные конфигурации линкера Cas12a-NLS в комплексе с каждой направляющей молекулой получали, по существу, как описано в Примере 2, с той лишь разницей, что эти комплексы собирали при двух концентрациях 20:60 и 80:240 пмоль Cas12a в качестве направляющей последовательности для каждой комбинации линкер Cas12a-NLS и направляющей молекулы. Комплексы направляющая Cas12a/нуклеопротеин трансфицировали в первичные Т-клетки как описано в Примере 3, и определяли конечную эффективность редактирования генома посредством комплексов направляющая Cas12a chRDNA/нуклеопротеин как описано в Примере 4.
[00557] Результаты редактирования конфигурации линкера Cas12a-NLS в комплексе с направляющей молекулой, показанной в Таблице 24, представлены на фиг. 16В и в Таблицах 25 и 26.
Активность редактирования конструкций Cas12a линкер-NLS для всех мишеней
Активность редактирования конструкций Cas12a линкер-NLS для всех мишеней
[00558] Данные, представленные на фиг. 16B и в Таблицах 25 и 26, продемонстрировали повышенную активность различных конфигураций NLS в отношении нескольких мишеней в первичных Т-клетках человека (см., например, средние показатели редактирования на фиг. 16B-1613 по сравнению со средними показателями редактирования на фиг. 16B-1616 или фиг. 16B- 1617). Альтернативные последовательности NLS, линкеры и нуклеазы Cas могут быть подвергнуты скринингу способом, аналогичным описанным здесь способам.
Пример 12
Реакция образования мультиплексных комплексов направляющая Cas12a chRDNA/нуклеопротеин
[00559] В этом примере описаны совместная доставка нескольких chRDNA Cas12a в процессе одной реакции трансфекции (реакции образования мультиплексных комплексов) в клетку и сравнение скоростей мультиплексного редактирования комплексами направляющая Cas12a chRDNA/нуклеопротеин с GS-SV40 (SEQ ID NO:479; «неоптимизированная NLS») и (G4S)2-NPL (SEQ ID NO:489; «оптимизированная NLS»).
[00560] A. Конструирование in silico последовательностей линкера Cas12a chRDNA-NLS
[00561] Белок Cas12a Acidaminococcus sp. (штамм BV3L6) (SEQ ID NO:01) был сконструирован либо с первой последовательностью С-концевой линкер-NLS (SEQ ID NO:479), либо со второй последовательностью С-концевой линкер-NLS (SEQ ID NO:489), и рекомбинантный белок Cas12a экспрессировали, как описано в Примере 2.
[00562] B. Активность образования мультиплексного комплекса с линкером Cas12a-NLS.
[00563] Очищенные рекомбинантные белки Cas12a, содержащие либо последовательность линкер-NLS SEQ ID NO:479, либо последовательность линкер-NLS SEQ ID NO:489, подвергали реакции образования комплекса либо с геном chRDNA, нацеленным на TRAC (SEQ ID NO:508), либо с геном chRDNA, нацеленным на B2M (SEQ ID NO:416), либо с геном chRDNA, нацеленным на CISH (SEQ ID NO:509), либо с геном chRDNA, нацеленным на CBLB (SEQ ID NO:510), как описано в Примере 2, с той лишь разницей, что эти комплексы были собраны в соотношении 40:80 пмоль Cas12a:направляющая молекула. Каждый комплекс направляющая Cas12a chRDNA/нуклеопротеин использовали как единый нацеливающий комплекс; комплексы направляющая Cas12a chRDNA/нуклеопротеин, нацеленные на TRAC и B2M, объединяли в одну смесь; комплексы направляющая Cas12a chRDNA/нуклеопротеин, нацеленные на CISH и CBLB, объединяли в одну смесь; или комплексы направляющая Cas12a chRDNA/нуклеопротеин, нацеленные на TRAC, B2M, CISH и CBLB, объединяли в одну смесь. Каждую композицию Cas12a chRDNA/нуклеопротеин переносили в первичные Т-клетки, как описано в Примере 3. Через сорок восемь часов после трансфекции определяли конечную эффективность редактирования генома посредством каждого комплекса Cas12a chRDNA/нуклеопротеин, как описано в Примере 4. Результаты эксперимента по редактированию в клетке показаны на фиг. 17 и представлены в Таблице 27.
Активность редактирования в клетке посредством мультиплексных конструкций линкер Cas12a-NLS
мишени
[00564] Данные, представленные на фиг. 17 и в Таблице 27, продемонстрировали повышенную активность конструкции линкер-NLS при ее использовании в реакции образования мультиплексных комплексов в первичных Т-клетках человека. См., например, среднее редактирование серий на фиг. 17, 1707 с неоптимизированным линкером GS-SV40-NLS (SEQ ID NO:479, фиг. 17, 1708) и среднее редактирование серий на Фиг. 17, 1711 с оптимизированным линкером (G4S)2-NPL-NLS (SEQ ID NO: 489, фиг. 17, 1712). Альтернативные последовательности NLS, линкеры, нуклеазы Cas12 и мишени для проведения мультиплексной реакции могут быть подвергнуты скринингу способом, аналогичным способам, описанным в настоящей заявке.
Пример 13
Редактирование с помощью комплексов направляющая Cas12a chRDNA/нуклеопротеин, включающих химические модификации
[00565] В этом примере описаны активности редактирования chRDNA Cas12a, содержащих фосфоротиоатные химические модификации в активирующей и нацеливающей области направляющей РНК Cas12a в клетках.
[00565] A. Конструирование chRDNA Cas12a in silico с химическими модификациями
[00567] Для конструирования была выбрана последовательность, нацеленная на TRAC-12 (SEQ ID NO:316). Две фосфортиоатные связи были введены у 5'-конца направляющей молекулы Cas12a (то есть, 5'-концевые нуклеотиды активирующей области), а две фосфортиоатные связи были введены у 3'-конца направляющей молекулы Cas12a (то есть, 3'-концевые нуклеотиды нацеливающей области). Затем была сконструирована серия направляющих последовательностей Cas12a с основаниями ДНК в активирующей области в дополнение к фосфоротиоатным модификациям, защищающим концевые участки. Последовательности Cas12a chRDNA с химическими модификациями показаны в Таблице 28. Последовательности были переданы коммерческому производителю для синтеза (перечисленные фосфоротиоатные связи и положения оснований ДНК соответствуют номерам, показанным на фиг. 5).
Cas12a chRDNA с химической модификацией
область
область
[00568] B. Трансфекция и анализ клеток
[00569] Отдельные комплексы направляющая Cas12a/нуклеопротеин получали, по существу, как описано в Примере 2. Комплексы нуклеопротеинов переносили в первичные Т-клетки, как описано в Примере 3, и конечную эффективность редактирования генома посредством комплексов направляющая Cas12a/нуклеопротеин определяли, как описано в Примере 4. Результаты эксперимента по редактированию в клетке показаны в Таблице 29.
Редактирование Cas12a chRDNA с химической модификацией в клетках
StDev=стандартное отклонение; n=2
[00570] Данные, представленные в Таблице 29, продемонстрировали активность редактирования направляющих молекул Cas12a, содержащих либо только фосфоротиоатные связи, либо фосфоротиоатные связи и основания ДНК. Эти направляющие молекулы обеспечивают надежное редактирование в первичных Т-клетках человека по сравнению со всеми направляющими РНК (см., например, Таблицу 29, то есть, где указано среднее редактирование SEQ ID NO:511 по сравнению со средним редактированием SEQ ID NO:512 или SEQ ID NO:515). Альтернативные комбинации и положения химической модификации могут быть подвергнуты скринингу способами, аналогичными описанным здесь способам.
Пример 14
Трансфекция человеческих индуцированных плюрипотентных стволовых клеток комплексами Cas12a chRDNA/нуклеопротеин
[00571] В этом Примере описано редактирование человеческих индуцированных плюрипотентных стволовых клеток (iPSC) посредством комплексов Cas12a chRDNA/нуклеопротеин.
[00572] A. Конструирование направляющих chRDNA in silico
[00573] Направляющая последовательность AsCas12a, нацеленная на ген CISH (SEQ ID NO:509) и содержащая основания ДНК в активирующей области направляющей молекулы, была отобрана для дальнейшего конструирования и введения дополнительных оснований ДНК в нацеливающую область (SEQ ID NO:518 - SEQ ID NO:529). Последовательности были переданы коммерческому производителю для синтеза.
[00574] B. Трансфекция и анализ клеток
[00575] Отдельные комплексы направляющая Cas12a/нуклеопротеин получали, по существу, как описано в примере 2. Нуклеопротеиновые комплексы переносили в первичные Т-клетки, как описано в Примере 3.
[00575] iPSC обрабатывали и трансфицировали способом, аналогичным способам обработки и трансфекции первичных Т-клеток, описанным в Примере 3, но со следующими модификациями. iPSC культивировали в среде mTeSR-plus (STEMCELL Technologies, Cambridge, MA), дополненной Rho-ассоциированным ингибитором протеинкиназы, содержащим суперспирализованную группу («ROCKi», MilliporeSigma, Burlington, МА) в конечной концентрации 10 мкМ в течение 3 часов при 37°C до трансфекции. Среду mTeSR-plus/ROCKi удаляли, iPSC промывали 10 мл PBS с последующим добавлением 3 мл аккутазы (STEMCELL Technologies, Cambridge, MA), и клетки инкубировали в течение 5-10 минут при 37°C. Затем к клеткам добавляли 7 мл mTeSR-импульса и ROCKi, после чего клетки перемешивали и подсчитывали. Затем клетки центрифугировали, среду удаляли, клетки промывали 10 мл PBS, снова центрифугировали и удаляли PBS. Клетки ресуспендировали в растворе Nucleofector™ P3 (Lonza, Allendale, NJ) до плотности 25 клеток/мл, смешанных с комплексами направляющая Cas12a chRDNA/нуклеопротеин и трансфицировали с использованием импульсного кода CA158. Полученные в результате эффективности редактирования генома посредством комплексов направляющая Cas12a/нуклеопротеин определяли как описано в Примере 4, и данные были представлены в Таблице 29.
Редактирование iPSC посредством Cas12a chRDNA
StDev=стандартное отклонение; n=1-2
[00577] Данные, представленные в Таблице 29, продемонстрировали, что комплексы направляющая Cas12a chRDNA/нуклеопротеин могут быть использованы для конструирования человеческих iPSC. Клетки других типов могут быть отредактированы способами, аналогичными описанным здесь способам.
[00578] Как очевидно для специалиста в данной области, модификации и изменения вышеприведенных вариантов осуществления изобретения могут быть введены так, чтобы они не выходили за рамки существа и объема настоящего изобретения. Такие модификации и изменения входят в объем настоящего изобретения.
--->
SEQUENCE LISTING
<110> CARIBOU BIOSCIENCES, INC.
<120> DNA-CONTAINING POLYNUCLEOTIDES AND GUIDES FOR CRISPR TYPE V
SYSTEMS, AND METHODS OF MAKING AND USING THE SAME
<130> CBI039.30
<140> PCT/US2021/055394
<141> 2021-10-18
<150> 63/229,870
<151> 2021-08-05
<150> 63/127,648
<151> 2020-12-18
<150> 63/093,459
<151> 2020-10-19
<160> 529
<170> PatentIn version 3.5
<210> 1
<211> 1307
<212> PRT
<213> Acidaminococcus sp.
<220>
<223> Acidaminococcus sp. (strain BV3L6)
<220>
<223> Cas12a
<400> 1
Met Thr Gln Phe Glu Gly Phe Thr Asn Leu Tyr Gln Val Ser Lys Thr
1 5 10 15
Leu Arg Phe Glu Leu Ile Pro Gln Gly Lys Thr Leu Lys His Ile Gln
20 25 30
Glu Gln Gly Phe Ile Glu Glu Asp Lys Ala Arg Asn Asp His Tyr Lys
35 40 45
Glu Leu Lys Pro Ile Ile Asp Arg Ile Tyr Lys Thr Tyr Ala Asp Gln
50 55 60
Cys Leu Gln Leu Val Gln Leu Asp Trp Glu Asn Leu Ser Ala Ala Ile
65 70 75 80
Asp Ser Tyr Arg Lys Glu Lys Thr Glu Glu Thr Arg Asn Ala Leu Ile
85 90 95
Glu Glu Gln Ala Thr Tyr Arg Asn Ala Ile His Asp Tyr Phe Ile Gly
100 105 110
Arg Thr Asp Asn Leu Thr Asp Ala Ile Asn Lys Arg His Ala Glu Ile
115 120 125
Tyr Lys Gly Leu Phe Lys Ala Glu Leu Phe Asn Gly Lys Val Leu Lys
130 135 140
Gln Leu Gly Thr Val Thr Thr Thr Glu His Glu Asn Ala Leu Leu Arg
145 150 155 160
Ser Phe Asp Lys Phe Thr Thr Tyr Phe Ser Gly Phe Tyr Glu Asn Arg
165 170 175
Lys Asn Val Phe Ser Ala Glu Asp Ile Ser Thr Ala Ile Pro His Arg
180 185 190
Ile Val Gln Asp Asn Phe Pro Lys Phe Lys Glu Asn Cys His Ile Phe
195 200 205
Thr Arg Leu Ile Thr Ala Val Pro Ser Leu Arg Glu His Phe Glu Asn
210 215 220
Val Lys Lys Ala Ile Gly Ile Phe Val Ser Thr Ser Ile Glu Glu Val
225 230 235 240
Phe Ser Phe Pro Phe Tyr Asn Gln Leu Leu Thr Gln Thr Gln Ile Asp
245 250 255
Leu Tyr Asn Gln Leu Leu Gly Gly Ile Ser Arg Glu Ala Gly Thr Glu
260 265 270
Lys Ile Lys Gly Leu Asn Glu Val Leu Asn Leu Ala Ile Gln Lys Asn
275 280 285
Asp Glu Thr Ala His Ile Ile Ala Ser Leu Pro His Arg Phe Ile Pro
290 295 300
Leu Phe Lys Gln Ile Leu Ser Asp Arg Asn Thr Leu Ser Phe Ile Leu
305 310 315 320
Glu Glu Phe Lys Ser Asp Glu Glu Val Ile Gln Ser Phe Cys Lys Tyr
325 330 335
Lys Thr Leu Leu Arg Asn Glu Asn Val Leu Glu Thr Ala Glu Ala Leu
340 345 350
Phe Asn Glu Leu Asn Ser Ile Asp Leu Thr His Ile Phe Ile Ser His
355 360 365
Lys Lys Leu Glu Thr Ile Ser Ser Ala Leu Cys Asp His Trp Asp Thr
370 375 380
Leu Arg Asn Ala Leu Tyr Glu Arg Arg Ile Ser Glu Leu Thr Gly Lys
385 390 395 400
Ile Thr Lys Ser Ala Lys Glu Lys Val Gln Arg Ser Leu Lys His Glu
405 410 415
Asp Ile Asn Leu Gln Glu Ile Ile Ser Ala Ala Gly Lys Glu Leu Ser
420 425 430
Glu Ala Phe Lys Gln Lys Thr Ser Glu Ile Leu Ser His Ala His Ala
435 440 445
Ala Leu Asp Gln Pro Leu Pro Thr Thr Leu Lys Lys Gln Glu Glu Lys
450 455 460
Glu Ile Leu Lys Ser Gln Leu Asp Ser Leu Leu Gly Leu Tyr His Leu
465 470 475 480
Leu Asp Trp Phe Ala Val Asp Glu Ser Asn Glu Val Asp Pro Glu Phe
485 490 495
Ser Ala Arg Leu Thr Gly Ile Lys Leu Glu Met Glu Pro Ser Leu Ser
500 505 510
Phe Tyr Asn Lys Ala Arg Asn Tyr Ala Thr Lys Lys Pro Tyr Ser Val
515 520 525
Glu Lys Phe Lys Leu Asn Phe Gln Met Pro Thr Leu Ala Ser Gly Trp
530 535 540
Asp Val Asn Lys Glu Lys Asn Asn Gly Ala Ile Leu Phe Val Lys Asn
545 550 555 560
Gly Leu Tyr Tyr Leu Gly Ile Met Pro Lys Gln Lys Gly Arg Tyr Lys
565 570 575
Ala Leu Ser Phe Glu Pro Thr Glu Lys Thr Ser Glu Gly Phe Asp Lys
580 585 590
Met Tyr Tyr Asp Tyr Phe Pro Asp Ala Ala Lys Met Ile Pro Lys Cys
595 600 605
Ser Thr Gln Leu Lys Ala Val Thr Ala His Phe Gln Thr His Thr Thr
610 615 620
Pro Ile Leu Leu Ser Asn Asn Phe Ile Glu Pro Leu Glu Ile Thr Lys
625 630 635 640
Glu Ile Tyr Asp Leu Asn Asn Pro Glu Lys Glu Pro Lys Lys Phe Gln
645 650 655
Thr Ala Tyr Ala Lys Lys Thr Gly Asp Gln Lys Gly Tyr Arg Glu Ala
660 665 670
Leu Cys Lys Trp Ile Asp Phe Thr Arg Asp Phe Leu Ser Lys Tyr Thr
675 680 685
Lys Thr Thr Ser Ile Asp Leu Ser Ser Leu Arg Pro Ser Ser Gln Tyr
690 695 700
Lys Asp Leu Gly Glu Tyr Tyr Ala Glu Leu Asn Pro Leu Leu Tyr His
705 710 715 720
Ile Ser Phe Gln Arg Ile Ala Glu Lys Glu Ile Met Asp Ala Val Glu
725 730 735
Thr Gly Lys Leu Tyr Leu Phe Gln Ile Tyr Asn Lys Asp Phe Ala Lys
740 745 750
Gly His His Gly Lys Pro Asn Leu His Thr Leu Tyr Trp Thr Gly Leu
755 760 765
Phe Ser Pro Glu Asn Leu Ala Lys Thr Ser Ile Lys Leu Asn Gly Gln
770 775 780
Ala Glu Leu Phe Tyr Arg Pro Lys Ser Arg Met Lys Arg Met Ala His
785 790 795 800
Arg Leu Gly Glu Lys Met Leu Asn Lys Lys Leu Lys Asp Gln Lys Thr
805 810 815
Pro Ile Pro Asp Thr Leu Tyr Gln Glu Leu Tyr Asp Tyr Val Asn His
820 825 830
Arg Leu Ser His Asp Leu Ser Asp Glu Ala Arg Ala Leu Leu Pro Asn
835 840 845
Val Ile Thr Lys Glu Val Ser His Glu Ile Ile Lys Asp Arg Arg Phe
850 855 860
Thr Ser Asp Lys Phe Phe Phe His Val Pro Ile Thr Leu Asn Tyr Gln
865 870 875 880
Ala Ala Asn Ser Pro Ser Lys Phe Asn Gln Arg Val Asn Ala Tyr Leu
885 890 895
Lys Glu His Pro Glu Thr Pro Ile Ile Gly Ile Asp Arg Gly Glu Arg
900 905 910
Asn Leu Ile Tyr Ile Thr Val Ile Asp Ser Thr Gly Lys Ile Leu Glu
915 920 925
Gln Arg Ser Leu Asn Thr Ile Gln Gln Phe Asp Tyr Gln Lys Lys Leu
930 935 940
Asp Asn Arg Glu Lys Glu Arg Val Ala Ala Arg Gln Ala Trp Ser Val
945 950 955 960
Val Gly Thr Ile Lys Asp Leu Lys Gln Gly Tyr Leu Ser Gln Val Ile
965 970 975
His Glu Ile Val Asp Leu Met Ile His Tyr Gln Ala Val Val Val Leu
980 985 990
Glu Asn Leu Asn Phe Gly Phe Lys Ser Lys Arg Thr Gly Ile Ala Glu
995 1000 1005
Lys Ala Val Tyr Gln Gln Phe Glu Lys Met Leu Ile Asp Lys Leu
1010 1015 1020
Asn Cys Leu Val Leu Lys Asp Tyr Pro Ala Glu Lys Val Gly Gly
1025 1030 1035
Val Leu Asn Pro Tyr Gln Leu Thr Asp Gln Phe Thr Ser Phe Ala
1040 1045 1050
Lys Met Gly Thr Gln Ser Gly Phe Leu Phe Tyr Val Pro Ala Pro
1055 1060 1065
Tyr Thr Ser Lys Ile Asp Pro Leu Thr Gly Phe Val Asp Pro Phe
1070 1075 1080
Val Trp Lys Thr Ile Lys Asn His Glu Ser Arg Lys His Phe Leu
1085 1090 1095
Glu Gly Phe Asp Phe Leu His Tyr Asp Val Lys Thr Gly Asp Phe
1100 1105 1110
Ile Leu His Phe Lys Met Asn Arg Asn Leu Ser Phe Gln Arg Gly
1115 1120 1125
Leu Pro Gly Phe Met Pro Ala Trp Asp Ile Val Phe Glu Lys Asn
1130 1135 1140
Glu Thr Gln Phe Asp Ala Lys Gly Thr Pro Phe Ile Ala Gly Lys
1145 1150 1155
Arg Ile Val Pro Val Ile Glu Asn His Arg Phe Thr Gly Arg Tyr
1160 1165 1170
Arg Asp Leu Tyr Pro Ala Asn Glu Leu Ile Ala Leu Leu Glu Glu
1175 1180 1185
Lys Gly Ile Val Phe Arg Asp Gly Ser Asn Ile Leu Pro Lys Leu
1190 1195 1200
Leu Glu Asn Asp Asp Ser His Ala Ile Asp Thr Met Val Ala Leu
1205 1210 1215
Ile Arg Ser Val Leu Gln Met Arg Asn Ser Asn Ala Ala Thr Gly
1220 1225 1230
Glu Asp Tyr Ile Asn Ser Pro Val Arg Asp Leu Asn Gly Val Cys
1235 1240 1245
Phe Asp Ser Arg Phe Gln Asn Pro Glu Trp Pro Met Asp Ala Asp
1250 1255 1260
Ala Asn Gly Ala Tyr His Ile Ala Leu Lys Gly Gln Leu Leu Leu
1265 1270 1275
Asn His Leu Lys Glu Ser Lys Asp Leu Lys Leu Gln Asn Gly Ile
1280 1285 1290
Ser Asn Gln Asp Trp Leu Ala Tyr Ile Gln Glu Leu Arg Asn
1295 1300 1305
<210> 2
<211> 1228
<212> PRT
<213> Unknown
<220>
<223> Description of Unknown:
Lachnospiraceae bacterium ND2006
<220>
<223> Cas12a
<400> 2
Met Ser Lys Leu Glu Lys Phe Thr Asn Cys Tyr Ser Leu Ser Lys Thr
1 5 10 15
Leu Arg Phe Lys Ala Ile Pro Val Gly Lys Thr Gln Glu Asn Ile Asp
20 25 30
Asn Lys Arg Leu Leu Val Glu Asp Glu Lys Arg Ala Glu Asp Tyr Lys
35 40 45
Gly Val Lys Lys Leu Leu Asp Arg Tyr Tyr Leu Ser Phe Ile Asn Asp
50 55 60
Val Leu His Ser Ile Lys Leu Lys Asn Leu Asn Asn Tyr Ile Ser Leu
65 70 75 80
Phe Arg Lys Lys Thr Arg Thr Glu Lys Glu Asn Lys Glu Leu Glu Asn
85 90 95
Leu Glu Ile Asn Leu Arg Lys Glu Ile Ala Lys Ala Phe Lys Gly Asn
100 105 110
Glu Gly Tyr Lys Ser Leu Phe Lys Lys Asp Ile Ile Glu Thr Ile Leu
115 120 125
Pro Glu Phe Leu Asp Asp Lys Asp Glu Ile Ala Leu Val Asn Ser Phe
130 135 140
Asn Gly Phe Thr Thr Ala Phe Thr Gly Phe Phe Asp Asn Arg Glu Asn
145 150 155 160
Met Phe Ser Glu Glu Ala Lys Ser Thr Ser Ile Ala Phe Arg Cys Ile
165 170 175
Asn Glu Asn Leu Thr Arg Tyr Ile Ser Asn Met Asp Ile Phe Glu Lys
180 185 190
Val Asp Ala Ile Phe Asp Lys His Glu Val Gln Glu Ile Lys Glu Lys
195 200 205
Ile Leu Asn Ser Asp Tyr Asp Val Glu Asp Phe Phe Glu Gly Glu Phe
210 215 220
Phe Asn Phe Val Leu Thr Gln Glu Gly Ile Asp Val Tyr Asn Ala Ile
225 230 235 240
Ile Gly Gly Phe Val Thr Glu Ser Gly Glu Lys Ile Lys Gly Leu Asn
245 250 255
Glu Tyr Ile Asn Leu Tyr Asn Gln Lys Thr Lys Gln Lys Leu Pro Lys
260 265 270
Phe Lys Pro Leu Tyr Lys Gln Val Leu Ser Asp Arg Glu Ser Leu Ser
275 280 285
Phe Tyr Gly Glu Gly Tyr Thr Ser Asp Glu Glu Val Leu Glu Val Phe
290 295 300
Arg Asn Thr Leu Asn Lys Asn Ser Glu Ile Phe Ser Ser Ile Lys Lys
305 310 315 320
Leu Glu Lys Leu Phe Lys Asn Phe Asp Glu Tyr Ser Ser Ala Gly Ile
325 330 335
Phe Val Lys Asn Gly Pro Ala Ile Ser Thr Ile Ser Lys Asp Ile Phe
340 345 350
Gly Glu Trp Asn Val Ile Arg Asp Lys Trp Asn Ala Glu Tyr Asp Asp
355 360 365
Ile His Leu Lys Lys Lys Ala Val Val Thr Glu Lys Tyr Glu Asp Asp
370 375 380
Arg Arg Lys Ser Phe Lys Lys Ile Gly Ser Phe Ser Leu Glu Gln Leu
385 390 395 400
Gln Glu Tyr Ala Asp Ala Asp Leu Ser Val Val Glu Lys Leu Lys Glu
405 410 415
Ile Ile Ile Gln Lys Val Asp Glu Ile Tyr Lys Val Tyr Gly Ser Ser
420 425 430
Glu Lys Leu Phe Asp Ala Asp Phe Val Leu Glu Lys Ser Leu Lys Lys
435 440 445
Asn Asp Ala Val Val Ala Ile Met Lys Asp Leu Leu Asp Ser Val Lys
450 455 460
Ser Phe Glu Asn Tyr Ile Lys Ala Phe Phe Gly Glu Gly Lys Glu Thr
465 470 475 480
Asn Arg Asp Glu Ser Phe Tyr Gly Asp Phe Val Leu Ala Tyr Asp Ile
485 490 495
Leu Leu Lys Val Asp His Ile Tyr Asp Ala Ile Arg Asn Tyr Val Thr
500 505 510
Gln Lys Pro Tyr Ser Lys Asp Lys Phe Lys Leu Tyr Phe Gln Asn Pro
515 520 525
Gln Phe Met Gly Gly Trp Asp Lys Asp Lys Glu Thr Asp Tyr Arg Ala
530 535 540
Thr Ile Leu Arg Tyr Gly Ser Lys Tyr Tyr Leu Ala Ile Met Asp Lys
545 550 555 560
Lys Tyr Ala Lys Cys Leu Gln Lys Ile Asp Lys Asp Asp Val Asn Gly
565 570 575
Asn Tyr Glu Lys Ile Asn Tyr Lys Leu Leu Pro Gly Pro Asn Lys Met
580 585 590
Leu Pro Lys Val Phe Phe Ser Lys Lys Trp Met Ala Tyr Tyr Asn Pro
595 600 605
Ser Glu Asp Ile Gln Lys Ile Tyr Lys Asn Gly Thr Phe Lys Lys Gly
610 615 620
Asp Met Phe Asn Leu Asn Asp Cys His Lys Leu Ile Asp Phe Phe Lys
625 630 635 640
Asp Ser Ile Ser Arg Tyr Pro Lys Trp Ser Asn Ala Tyr Asp Phe Asn
645 650 655
Phe Ser Glu Thr Glu Lys Tyr Lys Asp Ile Ala Gly Phe Tyr Arg Glu
660 665 670
Val Glu Glu Gln Gly Tyr Lys Val Ser Phe Glu Ser Ala Ser Lys Lys
675 680 685
Glu Val Asp Lys Leu Val Glu Glu Gly Lys Leu Tyr Met Phe Gln Ile
690 695 700
Tyr Asn Lys Asp Phe Ser Asp Lys Ser His Gly Thr Pro Asn Leu His
705 710 715 720
Thr Met Tyr Phe Lys Leu Leu Phe Asp Glu Asn Asn His Gly Gln Ile
725 730 735
Arg Leu Ser Gly Gly Ala Glu Leu Phe Met Arg Arg Ala Ser Leu Lys
740 745 750
Lys Glu Glu Leu Val Val His Pro Ala Asn Ser Pro Ile Ala Asn Lys
755 760 765
Asn Pro Asp Asn Pro Lys Lys Thr Thr Thr Leu Ser Tyr Asp Val Tyr
770 775 780
Lys Asp Lys Arg Phe Ser Glu Asp Gln Tyr Glu Leu His Ile Pro Ile
785 790 795 800
Ala Ile Asn Lys Cys Pro Lys Asn Ile Phe Lys Ile Asn Thr Glu Val
805 810 815
Arg Val Leu Leu Lys His Asp Asp Asn Pro Tyr Val Ile Gly Ile Asp
820 825 830
Arg Gly Glu Arg Asn Leu Leu Tyr Ile Val Val Val Asp Gly Lys Gly
835 840 845
Asn Ile Val Glu Gln Tyr Ser Leu Asn Glu Ile Ile Asn Asn Phe Asn
850 855 860
Gly Ile Arg Ile Lys Thr Asp Tyr His Ser Leu Leu Asp Lys Lys Glu
865 870 875 880
Lys Glu Arg Phe Glu Ala Arg Gln Asn Trp Thr Ser Ile Glu Asn Ile
885 890 895
Lys Glu Leu Lys Ala Gly Tyr Ile Ser Gln Val Val His Lys Ile Cys
900 905 910
Glu Leu Val Glu Lys Tyr Asp Ala Val Ile Ala Leu Glu Asp Leu Asn
915 920 925
Ser Gly Phe Lys Asn Ser Arg Val Lys Val Glu Lys Gln Val Tyr Gln
930 935 940
Lys Phe Glu Lys Met Leu Ile Asp Lys Leu Asn Tyr Met Val Asp Lys
945 950 955 960
Lys Ser Asn Pro Cys Ala Thr Gly Gly Ala Leu Lys Gly Tyr Gln Ile
965 970 975
Thr Asn Lys Phe Glu Ser Phe Lys Ser Met Ser Thr Gln Asn Gly Phe
980 985 990
Ile Phe Tyr Ile Pro Ala Trp Leu Thr Ser Lys Ile Asp Pro Ser Thr
995 1000 1005
Gly Phe Val Asn Leu Leu Lys Thr Lys Tyr Thr Ser Ile Ala Asp
1010 1015 1020
Ser Lys Lys Phe Ile Ser Ser Phe Asp Arg Ile Met Tyr Val Pro
1025 1030 1035
Glu Glu Asp Leu Phe Glu Phe Ala Leu Asp Tyr Lys Asn Phe Ser
1040 1045 1050
Arg Thr Asp Ala Asp Tyr Ile Lys Lys Trp Lys Leu Tyr Ser Tyr
1055 1060 1065
Gly Asn Arg Ile Arg Ile Phe Arg Asn Pro Lys Lys Asn Asn Val
1070 1075 1080
Phe Asp Trp Glu Glu Val Cys Leu Thr Ser Ala Tyr Lys Glu Leu
1085 1090 1095
Phe Asn Lys Tyr Gly Ile Asn Tyr Gln Gln Gly Asp Ile Arg Ala
1100 1105 1110
Leu Leu Cys Glu Gln Ser Asp Lys Ala Phe Tyr Ser Ser Phe Met
1115 1120 1125
Ala Leu Met Ser Leu Met Leu Gln Met Arg Asn Ser Ile Thr Gly
1130 1135 1140
Arg Thr Asp Val Asp Phe Leu Ile Ser Pro Val Lys Asn Ser Asp
1145 1150 1155
Gly Ile Phe Tyr Asp Ser Arg Asn Tyr Glu Ala Gln Glu Asn Ala
1160 1165 1170
Ile Leu Pro Lys Asn Ala Asp Ala Asn Gly Ala Tyr Asn Ile Ala
1175 1180 1185
Arg Lys Val Leu Trp Ala Ile Gly Gln Phe Lys Lys Ala Glu Asp
1190 1195 1200
Glu Lys Leu Asp Lys Val Lys Ile Ala Ile Ser Asn Lys Glu Trp
1205 1210 1215
Leu Glu Tyr Ala Gln Thr Ser Val Lys His
1220 1225
<210> 3
<211> 1300
<212> PRT
<213> Francisella tularensis
<220>
<223> Francisella tularensis subsp. novicida (strain U112)
<220>
<223> Cas12a
<400> 3
Met Ser Ile Tyr Gln Glu Phe Val Asn Lys Tyr Ser Leu Ser Lys Thr
1 5 10 15
Leu Arg Phe Glu Leu Ile Pro Gln Gly Lys Thr Leu Glu Asn Ile Lys
20 25 30
Ala Arg Gly Leu Ile Leu Asp Asp Glu Lys Arg Ala Lys Asp Tyr Lys
35 40 45
Lys Ala Lys Gln Ile Ile Asp Lys Tyr His Gln Phe Phe Ile Glu Glu
50 55 60
Ile Leu Ser Ser Val Cys Ile Ser Glu Asp Leu Leu Gln Asn Tyr Ser
65 70 75 80
Asp Val Tyr Phe Lys Leu Lys Lys Ser Asp Asp Asp Asn Leu Gln Lys
85 90 95
Asp Phe Lys Ser Ala Lys Asp Thr Ile Lys Lys Gln Ile Ser Glu Tyr
100 105 110
Ile Lys Asp Ser Glu Lys Phe Lys Asn Leu Phe Asn Gln Asn Leu Ile
115 120 125
Asp Ala Lys Lys Gly Gln Glu Ser Asp Leu Ile Leu Trp Leu Lys Gln
130 135 140
Ser Lys Asp Asn Gly Ile Glu Leu Phe Lys Ala Asn Ser Asp Ile Thr
145 150 155 160
Asp Ile Asp Glu Ala Leu Glu Ile Ile Lys Ser Phe Lys Gly Trp Thr
165 170 175
Thr Tyr Phe Lys Gly Phe His Glu Asn Arg Lys Asn Val Tyr Ser Ser
180 185 190
Asn Asp Ile Pro Thr Ser Ile Ile Tyr Arg Ile Val Asp Asp Asn Leu
195 200 205
Pro Lys Phe Leu Glu Asn Lys Ala Lys Tyr Glu Ser Leu Lys Asp Lys
210 215 220
Ala Pro Glu Ala Ile Asn Tyr Glu Gln Ile Lys Lys Asp Leu Ala Glu
225 230 235 240
Glu Leu Thr Phe Asp Ile Asp Tyr Lys Thr Ser Glu Val Asn Gln Arg
245 250 255
Val Phe Ser Leu Asp Glu Val Phe Glu Ile Ala Asn Phe Asn Asn Tyr
260 265 270
Leu Asn Gln Ser Gly Ile Thr Lys Phe Asn Thr Ile Ile Gly Gly Lys
275 280 285
Phe Val Asn Gly Glu Asn Thr Lys Arg Lys Gly Ile Asn Glu Tyr Ile
290 295 300
Asn Leu Tyr Ser Gln Gln Ile Asn Asp Lys Thr Leu Lys Lys Tyr Lys
305 310 315 320
Met Ser Val Leu Phe Lys Gln Ile Leu Ser Asp Thr Glu Ser Lys Ser
325 330 335
Phe Val Ile Asp Lys Leu Glu Asp Asp Ser Asp Val Val Thr Thr Met
340 345 350
Gln Ser Phe Tyr Glu Gln Ile Ala Ala Phe Lys Thr Val Glu Glu Lys
355 360 365
Ser Ile Lys Glu Thr Leu Ser Leu Leu Phe Asp Asp Leu Lys Ala Gln
370 375 380
Lys Leu Asp Leu Ser Lys Ile Tyr Phe Lys Asn Asp Lys Ser Leu Thr
385 390 395 400
Asp Leu Ser Gln Gln Val Phe Asp Asp Tyr Ser Val Ile Gly Thr Ala
405 410 415
Val Leu Glu Tyr Ile Thr Gln Gln Ile Ala Pro Lys Asn Leu Asp Asn
420 425 430
Pro Ser Lys Lys Glu Gln Glu Leu Ile Ala Lys Lys Thr Glu Lys Ala
435 440 445
Lys Tyr Leu Ser Leu Glu Thr Ile Lys Leu Ala Leu Glu Glu Phe Asn
450 455 460
Lys His Arg Asp Ile Asp Lys Gln Cys Arg Phe Glu Glu Ile Leu Ala
465 470 475 480
Asn Phe Ala Ala Ile Pro Met Ile Phe Asp Glu Ile Ala Gln Asn Lys
485 490 495
Asp Asn Leu Ala Gln Ile Ser Ile Lys Tyr Gln Asn Gln Gly Lys Lys
500 505 510
Asp Leu Leu Gln Ala Ser Ala Glu Asp Asp Val Lys Ala Ile Lys Asp
515 520 525
Leu Leu Asp Gln Thr Asn Asn Leu Leu His Lys Leu Lys Ile Phe His
530 535 540
Ile Ser Gln Ser Glu Asp Lys Ala Asn Ile Leu Asp Lys Asp Glu His
545 550 555 560
Phe Tyr Leu Val Phe Glu Glu Cys Tyr Phe Glu Leu Ala Asn Ile Val
565 570 575
Pro Leu Tyr Asn Lys Ile Arg Asn Tyr Ile Thr Gln Lys Pro Tyr Ser
580 585 590
Asp Glu Lys Phe Lys Leu Asn Phe Glu Asn Ser Thr Leu Ala Asn Gly
595 600 605
Trp Asp Lys Asn Lys Glu Pro Asp Asn Thr Ala Ile Leu Phe Ile Lys
610 615 620
Asp Asp Lys Tyr Tyr Leu Gly Val Met Asn Lys Lys Asn Asn Lys Ile
625 630 635 640
Phe Asp Asp Lys Ala Ile Lys Glu Asn Lys Gly Glu Gly Tyr Lys Lys
645 650 655
Ile Val Tyr Lys Leu Leu Pro Gly Ala Asn Lys Met Leu Pro Lys Val
660 665 670
Phe Phe Ser Ala Lys Ser Ile Lys Phe Tyr Asn Pro Ser Glu Asp Ile
675 680 685
Leu Arg Ile Arg Asn His Ser Thr His Thr Lys Asn Gly Ser Pro Gln
690 695 700
Lys Gly Tyr Glu Lys Phe Glu Phe Asn Ile Glu Asp Cys Arg Lys Phe
705 710 715 720
Ile Asp Phe Tyr Lys Gln Ser Ile Ser Lys His Pro Glu Trp Lys Asp
725 730 735
Phe Gly Phe Arg Phe Ser Asp Thr Gln Arg Tyr Asn Ser Ile Asp Glu
740 745 750
Phe Tyr Arg Glu Val Glu Asn Gln Gly Tyr Lys Leu Thr Phe Glu Asn
755 760 765
Ile Ser Glu Ser Tyr Ile Asp Ser Val Val Asn Gln Gly Lys Leu Tyr
770 775 780
Leu Phe Gln Ile Tyr Asn Lys Asp Phe Ser Ala Tyr Ser Lys Gly Arg
785 790 795 800
Pro Asn Leu His Thr Leu Tyr Trp Lys Ala Leu Phe Asp Glu Arg Asn
805 810 815
Leu Gln Asp Val Val Tyr Lys Leu Asn Gly Glu Ala Glu Leu Phe Tyr
820 825 830
Arg Lys Gln Ser Ile Pro Lys Lys Ile Thr His Pro Ala Lys Glu Ala
835 840 845
Ile Ala Asn Lys Asn Lys Asp Asn Pro Lys Lys Glu Ser Val Phe Glu
850 855 860
Tyr Asp Leu Ile Lys Asp Lys Arg Phe Thr Glu Asp Lys Phe Phe Phe
865 870 875 880
His Cys Pro Ile Thr Ile Asn Phe Lys Ser Ser Gly Ala Asn Lys Phe
885 890 895
Asn Asp Glu Ile Asn Leu Leu Leu Lys Glu Lys Ala Asn Asp Val His
900 905 910
Ile Leu Ser Ile Asp Arg Gly Glu Arg His Leu Ala Tyr Tyr Thr Leu
915 920 925
Val Asp Gly Lys Gly Asn Ile Ile Lys Gln Asp Thr Phe Asn Ile Ile
930 935 940
Gly Asn Asp Arg Met Lys Thr Asn Tyr His Asp Lys Leu Ala Ala Ile
945 950 955 960
Glu Lys Asp Arg Asp Ser Ala Arg Lys Asp Trp Lys Lys Ile Asn Asn
965 970 975
Ile Lys Glu Met Lys Glu Gly Tyr Leu Ser Gln Val Val His Glu Ile
980 985 990
Ala Lys Leu Val Ile Glu Tyr Asn Ala Ile Val Val Phe Glu Asp Leu
995 1000 1005
Asn Phe Gly Phe Lys Arg Gly Arg Phe Lys Val Glu Lys Gln Val
1010 1015 1020
Tyr Gln Lys Leu Glu Lys Met Leu Ile Glu Lys Leu Asn Tyr Leu
1025 1030 1035
Val Phe Lys Asp Asn Glu Phe Asp Lys Thr Gly Gly Val Leu Arg
1040 1045 1050
Ala Tyr Gln Leu Thr Ala Pro Phe Glu Thr Phe Lys Lys Met Gly
1055 1060 1065
Lys Gln Thr Gly Ile Ile Tyr Tyr Val Pro Ala Gly Phe Thr Ser
1070 1075 1080
Lys Ile Cys Pro Val Thr Gly Phe Val Asn Gln Leu Tyr Pro Lys
1085 1090 1095
Tyr Glu Ser Val Ser Lys Ser Gln Glu Phe Phe Ser Lys Phe Asp
1100 1105 1110
Lys Ile Cys Tyr Asn Leu Asp Lys Gly Tyr Phe Glu Phe Ser Phe
1115 1120 1125
Asp Tyr Lys Asn Phe Gly Asp Lys Ala Ala Lys Gly Lys Trp Thr
1130 1135 1140
Ile Ala Ser Phe Gly Ser Arg Leu Ile Asn Phe Arg Asn Ser Asp
1145 1150 1155
Lys Asn His Asn Trp Asp Thr Arg Glu Val Tyr Pro Thr Lys Glu
1160 1165 1170
Leu Glu Lys Leu Leu Lys Asp Tyr Ser Ile Glu Tyr Gly His Gly
1175 1180 1185
Glu Cys Ile Lys Ala Ala Ile Cys Gly Glu Ser Asp Lys Lys Phe
1190 1195 1200
Phe Ala Lys Leu Thr Ser Val Leu Asn Thr Ile Leu Gln Met Arg
1205 1210 1215
Asn Ser Lys Thr Gly Thr Glu Leu Asp Tyr Leu Ile Ser Pro Val
1220 1225 1230
Ala Asp Val Asn Gly Asn Phe Phe Asp Ser Arg Gln Ala Pro Lys
1235 1240 1245
Asn Met Pro Gln Asp Ala Asp Ala Asn Gly Ala Tyr His Ile Gly
1250 1255 1260
Leu Lys Gly Leu Met Leu Leu Gly Arg Ile Lys Asn Asn Gln Glu
1265 1270 1275
Gly Lys Lys Leu Asn Leu Val Ile Lys Asn Glu Glu Tyr Phe Glu
1280 1285 1290
Phe Val Gln Asn Arg Asn Met
1295 1300
<210> 4
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<220>
<223> Nuclear localization sequence (NLS)
<400> 4
Pro Lys Lys Lys Arg Lys Val
1 5
<210> 5
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<220>
<223> Nuclear localization sequence (NLS)
<400> 5
Lys Arg Pro Ala Ala Thr Lys Lys Ala Gly Gln Ala Lys Lys Lys Lys
1 5 10 15
<210> 6
<211> 20
<212> RNA
<213> Acidaminococcus sp.
<220>
<223> Acidaminococcus sp. (strain BV3L6)
<220>
<223> AsCas12 CRISPR repeat
<400> 6
uaauuucuac ucuuguagau 20
<210> 7
<211> 40
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> AsCas12 crRNA
<220>
<221> modified_base
<222> (21)..(40)
<223> a, c, g, or u
<400> 7
uaauuucuac ucuuguagau nnnnnnnnnn nnnnnnnnnn 40
<210> 8
<211> 21
<212> RNA
<213> Unknown
<220>
<223> Description of Unknown:
Lachnospiraceae bacterium ND2006
<220>
<223> LbaCas12 CRISPR repeat
<400> 8
uaauuucuac uaaguguaga u 21
<210> 9
<211> 41
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> LbaCas12 crRNA
<220>
<221> modified_base
<222> (22)..(41)
<223> a, c, g, or u
<400> 9
uaauuucuac uaaguguaga unnnnnnnnn nnnnnnnnnn n 41
<210> 10
<211> 20
<212> RNA
<213> Francisella tularensis
<220>
<223> Francisella tularensis subsp. novicida (strain U112)
<220>
<223> FnoCas12 CRISPR repeat
<400> 10
uaauuucuac uguuguagau 20
<210> 11
<211> 40
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> FnoCas12 crRNA
<220>
<221> modified_base
<222> (21)..(40)
<223> a, c, g, or u
<400> 11
uaauuucuac uguuguagau nnnnnnnnnn nnnnnnnnnn 40
<210> 12
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> TRAC-tgt1
<400> 12
tttctctcct ctttcccaca gaat 24
<210> 13
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> TRAC-tgt2
<400> 13
tttcccacag aatttagcat ggct 24
<210> 14
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> TRAC-tgt3
<400> 14
tttagcatgg ctcagacagt cact 24
<210> 15
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> TRAC-tgt4
<400> 15
tttattctgg tacaagcagc ctcc 24
<210> 16
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> TRAC-tgt5
<400> 16
tttctggaag ttcacagaga aacg 24
<210> 17
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> TRAC-tgt6
<400> 17
tttctctgtg aacttccaga aagc 24
<210> 18
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> TRAC-tgt7
<400> 18
tttggctgct ttctggaagt tcac 24
<210> 19
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> TRAC-tgt8
<400> 19
tttatcaacc tcaccaactc acac 24
<210> 20
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> TRAC-tgt9
<400> 20
tttgctgtca gtccaagctc catg 24
<210> 21
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> TRAC-tgt10
<400> 21
tttccaaaga caagaggtgt gttt 24
<210> 22
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> TRAC-tgt11
<400> 22
tttggaaagg gcacaagact ttct 24
<210> 23
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> TRAC-tgt12
<400> 23
tttagagtct ctcagctggt acac 24
<210> 24
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> TRAC-tgt13
<400> 24
tttgagaatc aaaatcggtg aata 24
<210> 25
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> TRAC-tgt14
<400> 25
tttgtttgag aatcaaaatc ggtg 24
<210> 26
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> TRAC-tgt15
<400> 26
tttgattctc aaacaaatgt gtca 24
<210> 27
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> TRAC-tgt16
<400> 27
tttgtgacac atttgtttga gaat 24
<210> 28
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> TRAC-tgt17
<400> 28
tttgtctgtg atatacacat caga 24
<210> 29
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> TRAC-tgt18
<400> 29
tttgttgctc caggccacag cact 24
<210> 30
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> TRAC-tgt19
<400> 30
tttgcacatg caaagtcaga tttg 24
<210> 31
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> TRAC-tgt20
<400> 31
tttgcatgtg caaacgcctt caac 24
<210> 32
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> TRAC-tgt21
<400> 32
tttcctttta gaaagttcct gtga 24
<210> 33
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> TRAC-tgt22
<400> 33
tttagaaagt tcctgtgatg tcaa 24
<210> 34
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> TRAC-tgt23
<400> 34
tttctcgacc agcttgacat caca 24
<210> 35
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> TRAC-tgt24
<400> 35
tttcaaagct tttctcgacc agct 24
<210> 36
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> TRAC-tgt25
<400> 36
tttacagata cgaacctaaa cttt 24
<210> 37
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> TRAC-tgt26
<400> 37
tttgaaagtt taggttcgta tctg 24
<210> 38
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> TRAC-tgt27
<400> 38
tttcaaaacc tgtcagtgat tggg 24
<210> 39
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> TRAC-tgt28
<400> 39
tttcaggagg aggattcgga accc 24
<210> 40
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> TRAC-tgt29
<400> 40
tttaatctgc tcatgacgct gcgg 24
<210> 41
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> TRAC-tgt30
<400> 41
tttggagagg gagaagaggg gcaa 24
<210> 42
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> TRAC-tgt31
<400> 42
tttcacctcc ttgggggtag gaga 24
<210> 43
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> TRAC-tgt32
<400> 43
tttatgatta agattgctga agag 24
<210> 44
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> TRAC-tgt33
<400> 44
tttggcagct cttcagcaat ctta 24
<210> 45
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> TRAC-tgt34
<400> 45
tttatttttt tttaatagtg ttca 24
<210> 46
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> TRAC-tgt35
<400> 46
tttatgaaca ctattaaaaa aaaa 24
<210> 47
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> TRAC-tgt36
<400> 47
tttctttatg aacactatta aaaa 24
<210> 48
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> TRAC-tgt37
<400> 48
tttaatagtg ttcataaaga aata 24
<210> 49
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> TRAC-tgt38
<400> 49
tttcccccca cgtcttgaga agaa 24
<210> 50
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> TRAC-tgt39
<400> 50
tttaatgaag gcatcggcag cagg 24
<210> 51
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt1
<400> 51
tttaatataa gtggaggcgt cgcg 24
<210> 52
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt2
<400> 52
tttctggcct ggaggctatc cagc 24
<210> 53
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt3
<400> 53
tttcccgata ttcctcaggt actc 24
<210> 54
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt4
<400> 54
tttactcacg tcatccagca gaga 24
<210> 55
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt5
<400> 55
tttccattct ctgctggatg acgt 24
<210> 56
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt6
<400> 56
tttgactttc cattctctgc tgga 24
<210> 57
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt7
<400> 57
tttcctgaat tgctatgtgt ctgg 24
<210> 58
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt8
<400> 58
tttcatccat ccgacattga agtt 24
<210> 59
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt9
<400> 59
tttcaattct ctctccattc ttca 24
<210> 60
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt10
<400> 60
tttcagcaag gactggtctt tcta 24
<210> 61
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt11
<400> 61
tttctatctc ttgtactaca ctga 24
<210> 62
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt12
<400> 62
tttcagtggg ggtgaattca gtgt 24
<210> 63
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt13
<400> 63
tttgtcacag cccaagatag ttaa 24
<210> 64
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt14
<400> 64
tttctccact gtctttttca taga 24
<210> 65
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt15
<400> 65
tttcatagat cgagacatgt aagc 24
<210> 66
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt16
<400> 66
tttctcaagg tcaaaaactt acct 24
<210> 67
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt17
<400> 67
tttcttttct tttcaggttt gaag 24
<210> 68
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt18
<400> 68
tttcttttca ggtttgaaga tgcc 24
<210> 69
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt19
<400> 69
tttcaggttt gaagatgccg catt 24
<210> 70
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt20
<400> 70
tttgaagatg ccgcatttgg attg 24
<210> 71
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt21
<400> 71
tttggaattc atccaatcca aatg 24
<210> 72
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt22
<400> 72
tttggattgg atgaattcca aatt 24
<210> 73
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt23
<400> 73
tttaatattg atatgcttat acac 24
<210> 74
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt24
<400> 74
tttgtgcata aagtgtaagt gtat 24
<210> 75
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt25
<400> 75
tttatgcaca aaatgtaggg ttat 24
<210> 76
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt26
<400> 76
tttataattc tactttgagt gctg 24
<210> 77
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt27
<400> 77
tttgagtgct gtctccatgt ttga 24
<210> 78
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt28
<400> 78
tttgatgtat ctgagcaggt tgct 24
<210> 79
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt29
<400> 79
tttgaactct tcaatctctt gcac 24
<210> 80
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt30
<400> 80
tttgagtgca agagattgaa gagt 24
<210> 81
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt31
<400> 81
tttacatact ctgcttagaa tttg 24
<210> 82
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt32
<400> 82
tttcccccaa attctaagca gagt 24
<210> 83
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt33
<400> 83
tttctaaatt ttcccccaaa ttct 24
<210> 84
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt34
<400> 84
tttgggggaa aatttagaaa tata 24
<210> 85
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt35
<400> 85
tttagaaata taattgacag gatt 24
<210> 86
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt36
<400> 86
tttccaataa tcctgtcaat tata 24
<210> 87
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt37
<400> 87
tttcattcat tataacaaat ttcc 24
<210> 88
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt38
<400> 88
tttgttataa tgaatgaaac attt 24
<210> 89
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt39
<400> 89
tttgtcatat aagattcata ttta 24
<210> 90
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt40
<400> 90
tttacttctt atacatttga taaa 24
<210> 91
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt41
<400> 91
tttatcaaat gtataagaag taaa 24
<210> 92
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt42
<400> 92
tttgataaag taaggcatgg ttgt 24
<210> 93
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt43
<400> 93
tttatttttg ttccacaagt taaa 24
<210> 94
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt44
<400> 94
tttaacttgt ggaacaaaaa taaa 24
<210> 95
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt45
<400> 95
tttatttaac ttgtggaaca aaaa 24
<210> 96
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt46
<400> 96
tttgttccac aagttaaata aatc 24
<210> 97
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt47
<400> 97
tttatgattt atttaacttg tgga 24
<210> 98
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt48
<400> 98
tttgaaaata aaggggtaat agtg 24
<210> 99
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt49
<400> 99
tttccctgtt tgaaaataaa gggg 24
<210> 100
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt50
<400> 100
tttattttca aacagggaaa cagt 24
<210> 101
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt51
<400> 101
tttcaaacag ggaaacagtc ttca 24
<210> 102
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt52
<400> 102
tttaccaagt ggaacttgaa gact 24
<210> 103
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt53
<400> 103
tttacactgt gagccaaact ctat 24
<210> 104
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt54
<400> 104
tttggctcac agtgtaaagg gcct 24
<210> 105
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt55
<400> 105
tttccagatt aggaatctga tgct 24
<210> 106
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt56
<400> 106
tttgagcatc agattcctaa tctg 24
<210> 107
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt57
<400> 107
tttaacttct ttgagcatca gatt 24
<210> 108
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt58
<400> 108
tttagccagc gttctttcct gcgg 24
<210> 109
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt59
<400> 109
tttcctgcgg gccaggtcat gagg 24
<210> 110
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt60
<400> 110
tttgctccct cttagagtct gcat 24
<210> 111
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt61
<400> 111
tttaatattt tagcaaggaa taga 24
<210> 112
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt62
<400> 112
tttagcaagg aatagatata caat 24
<210> 113
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt63
<400> 113
tttcaggacc ccttcttgga cacc 24
<210> 114
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt64
<400> 114
tttggtgtcc aagaaggggt cctg 24
<210> 115
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt65
<400> 115
tttatagaag ggacttaaat atcc 24
<210> 116
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt66
<400> 116
tttaagtccc ttctataaaa tggt 24
<210> 117
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt67
<400> 117
tttgcatata acctatccac atcc 24
<210> 118
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt68
<400> 118
tttaaagtat acaggaggat gtgg 24
<210> 119
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt69
<400> 119
tttaaatcat ttctagatta cttg 24
<210> 120
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt70
<400> 120
tttctagatt acttgtaata ccta 24
<210> 121
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt71
<400> 121
tttacattgt attaggtatt acaa 24
<210> 122
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt72
<400> 122
tttgcatagc atttacattg tatt 24
<210> 123
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt73
<400> 123
tttcttgtca ttattcctta aaca 24
<210> 124
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt74
<400> 124
tttaaggaat aatgacaaga aaaa 24
<210> 125
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt75
<400> 125
tttactgagc atgtacagac tttt 24
<210> 126
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt76
<400> 126
tttccccagt gtttttgatc catg 24
<210> 127
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt77
<400> 127
tttgatccat ggtttgctga atcc 24
<210> 128
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt78
<400> 128
tttgctgaat ccacagatgt ggag 24
<210> 129
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt79
<400> 129
tttgtcattc aaagtacagc gggc 24
<210> 130
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt80
<400> 130
tttgaatgac aaataacaga ttta 24
<210> 131
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt81
<400> 131
tttaaatctg ttatttgtca ttca 24
<210> 132
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt82
<400> 132
tttaaaattt tcaaggcata gttt 24
<210> 133
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> B2M-tgt83
<400> 133
tttcaaggca tagttttata cctg 24
<210> 134
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> PDCD1-tgt1
<400> 134
tttaattata attataatat aata 24
<210> 135
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> PDCD1-tgt2
<400> 135
tttaaagggg ttggccgggc tccc 24
<210> 136
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> PDCD1-tgt3
<400> 136
tttaaataat ttcaggaatg ggtt 24
<210> 137
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> PDCD1-tgt4
<400> 137
tttcaggaat gggttccaag gaga 24
<210> 138
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> PDCD1-tgt5
<400> 138
tttcaggccc agccagcact ctgg 24
<210> 139
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> PDCD1-tgt6
<400> 139
tttgtggggc agggaagctg aggc 24
<210> 140
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> PDCD1-tgt7
<400> 140
tttacacatg cccaggcagc acct 24
<210> 141
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> PDCD1-tgt8
<400> 141
tttcctgccc tgcccaccac agcc 24
<210> 142
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> PDCD1-tgt9
<400> 142
tttcagggaa ggtcagaaga gctc 24
<210> 143
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> PDCD1-tgt10
<400> 143
tttcgggatg ccactgccag gggc 24
<210> 144
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> PDCD1-tgt11
<400> 144
tttcctcagg agaagcaggc aggg 24
<210> 145
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> PDCD1-tgt12
<400> 145
tttcctagcg gaatgggcac ctca 24
<210> 146
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> PDCD1-tgt13
<400> 146
tttccagtgg cgagagaaga cccc 24
<210> 147
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> PDCD1-tgt14
<400> 147
tttctcctca aagaaggagg accc 24
<210> 148
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> PDCD1-tgt15
<400> 148
tttctctgca gggacaatag gagc 24
<210> 149
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> PDCD1-tgt16
<400> 149
tttggaactg gccggctggc ctgg 24
<210> 150
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> PDCD1-tgt17
<400> 150
tttgtgccct tccagagaga aggg 24
<210> 151
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> PDCD1-tgt18
<400> 151
tttgatctgc gccttggggg ccag 24
<210> 152
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> PDCD1-tgt19
<400> 152
tttagcacga agctctccga tgtg 24
<210> 153
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> PDCD1-tgt20
<400> 153
tttcccttcc gctcacctcc gcct 24
<210> 154
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> PDCD1-tgt21
<400> 154
cttactgcct cagcttccct gccc 24
<210> 155
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> PDCD1-tgt22
<400> 155
cttagcatgc tctcatattt aatt 24
<210> 156
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> CISH-tgt1
<400> 156
tttaccctag gtgagcaccc cctt 24
<210> 157
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> CISH-tgt2
<400> 157
tttgttccct gacacccaac agta 24
<210> 158
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> CISH-tgt3
<400> 158
tttagactgc tgcgctccaa agcg 24
<210> 159
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> CISH-tgt4
<400> 159
tttggagcgc agcagtctaa aact 24
<210> 160
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> CISH-tgt5
<400> 160
tttgtacgca gaagcagtgc ccgc 24
<210> 161
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> CISH-tgt6
<400> 161
tttaggtgta cagcagtggc tggt 24
<210> 162
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> CISH-tgt7
<400> 162
tttccggatg tggtcagcct tgtg 24
<210> 163
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> CISH-tgt8
<400> 163
tttcactgac agcgtgaaca ggta 24
<210> 164
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> CISH-tgt9
<400> 164
tttggctcac tctctgtctg ggct 24
<210> 165
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> CISH-tgt10
<400> 165
tttgctggct gtggagcgga ctgg 24
<210> 166
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> CBLB-tgt1
<400> 166
tttctgccat tcattgagtt tgcc 24
<210> 167
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> CBLB-tgt2
<400> 167
tttcctcctc gaccaccagg gttt 24
<210> 168
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> CBLB-tgt3
<400> 168
tttcggggat ttcctcctcg acca 24
<210> 169
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> CBLB-tgt4
<400> 169
tttgggtatt attgatgcta ttca 24
<210> 170
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> CBLB-tgt5
<400> 170
tttgggattt tggcacagtc ttac 24
<210> 171
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> CBLB-tgt6
<400> 171
tttgcctgat acatatcagc attt 24
<210> 172
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> CBLB-tgt7
<400> 172
tttaaagtac tcattctcac tgag 24
<210> 173
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> CBLB-tgt8
<400> 173
tttgactttt tcataaggct atca 24
<210> 174
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> CBLB-tgt9
<400> 174
tttaaagagt cttattgccc gttt 24
<210> 175
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> CBLB-tgt10
<400> 175
tttcttagct gtgacacatc cagg 24
<210> 176
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> CBLB-tgt11
<400> 176
tttctgtagt cgtgctttaa cttc 24
<210> 177
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> CBLB-tgt12
<400> 177
tttggtgcta tatttctgta gtcg 24
<210> 178
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> CBLB-tgt13
<400> 178
tttcacaata taattcatat tgtt 24
<210> 179
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> CBLB-tgt14
<400> 179
tttgtcattc tctgcacaaa tctt 24
<210> 180
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> CBLB-tgt15
<400> 180
tttcagctct gtaagatttg tgca 24
<210> 181
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> CBLB-tgt16
<400> 181
tttgtgcaga gaatgacaaa gatg 24
<210> 182
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> CBLB-tgt17
<400> 182
tttgatgtgc acctcttgcc ttac 24
<210> 183
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> CBLB-tgt17
<400> 183
tttgatgtgc acctcttgcc ttac 24
<210> 184
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> CBLB-tgt17
<400> 184
tttgatgtgc acctcttgcc ttac 24
<210> 185
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> CBLB-tgt20
<400> 185
tttctgtcgt tgtgaaataa aagg 24
<210> 186
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> CBLB-tgt21
<400> 186
tttcaggtgg cggtggagga ggat 24
<210> 187
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> CBLB-tgt22
<400> 187
tttccacatg atggatgtgt ctac 24
<210> 188
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> CBLB-tgt23
<400> 188
tttgggacta atcagcttgt ggga 24
<210> 189
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> CBLB-tgt24
<400> 189
tttgaactcg ctgtgattcc aggt 24
<210> 190
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> crRNA
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, g, or u
<400> 190
nnnnnnnnnn nnnnnnnnnn 20
<210> 191
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> d1
<220>
<221> modified_base
<222> (1)..(1)
<223> a, c, g, or t
<220>
<221> modified_base
<222> (2)..(20)
<223> a, c, g, or u
<400> 191
nnnnnnnnnn nnnnnnnnnn 20
<210> 192
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> d2
<220>
<221> modified_base
<222> (1)..(1)
<223> a, c, g, or u
<220>
<221> modified_base
<222> (2)..(2)
<223> a, c, g, or t
<220>
<221> modified_base
<222> (3)..(20)
<223> a, c, g, or u
<400> 192
nnnnnnnnnn nnnnnnnnnn 20
<210> 193
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> d3
<220>
<221> modified_base
<222> (1)..(2)
<223> a, c, g, or u
<220>
<221> modified_base
<222> (3)..(3)
<223> a, c, g, or t
<220>
<221> modified_base
<222> (4)..(20)
<223> a, c, g, or u
<400> 193
nnnnnnnnnn nnnnnnnnnn 20
<210> 194
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> d4
<220>
<221> modified_base
<222> (1)..(3)
<223> a, c, g, or u
<220>
<221> modified_base
<222> (4)..(4)
<223> a, c, g, or t
<220>
<221> modified_base
<222> (5)..(20)
<223> a, c, g, or u
<400> 194
nnnnnnnnnn nnnnnnnnnn 20
<210> 195
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> d5
<220>
<221> modified_base
<222> (1)..(4)
<223> a, c, g, or u
<220>
<221> modified_base
<222> (5)..(5)
<223> a, c, g, or t
<220>
<221> modified_base
<222> (6)..(20)
<223> a, c, g, or u
<400> 195
nnnnnnnnnn nnnnnnnnnn 20
<210> 196
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> d6
<220>
<221> modified_base
<222> (1)..(5)
<223> a, c, g, or u
<220>
<221> modified_base
<222> (6)..(6)
<223> a, c, g, or t
<220>
<221> modified_base
<222> (7)..(20)
<223> a, c, g, or u
<400> 196
nnnnnnnnnn nnnnnnnnnn 20
<210> 197
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> d7
<220>
<221> modified_base
<222> (1)..(6)
<223> a, c, g, or u
<220>
<221> modified_base
<222> (7)..(7)
<223> a, c, g, or t
<220>
<221> modified_base
<222> (8)..(20)
<223> a, c, g, or u
<400> 197
nnnnnnnnnn nnnnnnnnnn 20
<210> 198
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> d8
<220>
<221> modified_base
<222> (1)..(7)
<223> a, c, g, or u
<220>
<221> modified_base
<222> (8)..(8)
<223> a, c, g, or t
<220>
<221> modified_base
<222> (9)..(20)
<223> a, c, g, or u
<400> 198
nnnnnnnnnn nnnnnnnnnn 20
<210> 199
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> d9
<220>
<221> modified_base
<222> (1)..(8)
<223> a, c, g, or u
<220>
<221> modified_base
<222> (9)..(9)
<223> a, c, g, or t
<220>
<221> modified_base
<222> (10)..(20)
<223> a, c, g, or u
<400> 199
nnnnnnnnnn nnnnnnnnnn 20
<210> 200
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> d10
<220>
<221> modified_base
<222> (1)..(9)
<223> a, c, g, or u
<220>
<221> modified_base
<222> (10)..(10)
<223> a, c, g, or t
<220>
<221> modified_base
<222> (11)..(20)
<223> a, c, g, or u
<400> 200
nnnnnnnnnn nnnnnnnnnn 20
<210> 201
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> d11
<220>
<221> modified_base
<222> (1)..(10)
<223> a, c, g, or u
<220>
<221> modified_base
<222> (11)..(11)
<223> a, c, g, or t
<220>
<221> modified_base
<222> (12)..(20)
<223> a, c, g, or u
<400> 201
nnnnnnnnnn nnnnnnnnnn 20
<210> 202
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> d12
<220>
<221> modified_base
<222> (1)..(11)
<223> a, c, g, or u
<220>
<221> modified_base
<222> (12)..(12)
<223> a, c, g, or t
<220>
<221> modified_base
<222> (13)..(20)
<223> a, c, g, or u
<400> 202
nnnnnnnnnn nnnnnnnnnn 20
<210> 203
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> d13
<220>
<221> modified_base
<222> (1)..(12)
<223> a, c, g, or u
<220>
<221> modified_base
<222> (13)..(13)
<223> a, c, g, or t
<220>
<221> modified_base
<222> (14)..(20)
<223> a, c, g, or u
<400> 203
nnnnnnnnnn nnnnnnnnnn 20
<210> 204
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> d14
<220>
<221> modified_base
<222> (1)..(13)
<223> a, c, g, or u
<220>
<221> modified_base
<222> (14)..(14)
<223> a, c, g, or t
<220>
<221> modified_base
<222> (15)..(20)
<223> a, c, g, or u
<400> 204
nnnnnnnnnn nnnnnnnnnn 20
<210> 205
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> d15
<220>
<221> modified_base
<222> (1)..(14)
<223> a, c, g, or u
<220>
<221> modified_base
<222> (15)..(15)
<223> a, c, g, or t
<220>
<221> modified_base
<222> (16)..(20)
<223> a, c, g, or u
<400> 205
nnnnnnnnnn nnnnnnnnnn 20
<210> 206
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> d16
<220>
<221> modified_base
<222> (1)..(15)
<223> a, c, g, or u
<220>
<221> modified_base
<222> (16)..(16)
<223> a, c, g, or t
<220>
<221> modified_base
<222> (17)..(20)
<223> a, c, g, or u
<400> 206
nnnnnnnnnn nnnnnnnnnn 20
<210> 207
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> d17
<220>
<221> modified_base
<222> (1)..(16)
<223> a, c, g, or u
<220>
<221> modified_base
<222> (17)..(17)
<223> a, c, g, or t
<220>
<221> modified_base
<222> (18)..(20)
<223> a, c, g, or u
<400> 207
nnnnnnnnnn nnnnnnnnnn 20
<210> 208
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> d18
<220>
<221> modified_base
<222> (1)..(17)
<223> a, c, g, or u
<220>
<221> modified_base
<222> (18)..(18)
<223> a, c, g, or t
<220>
<221> modified_base
<222> (19)..(20)
<223> a, c, g, or u
<400> 208
nnnnnnnnnn nnnnnnnnnn 20
<210> 209
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> d19
<220>
<221> modified_base
<222> (1)..(18)
<223> a, c, g, or u
<220>
<221> modified_base
<222> (19)..(19)
<223> a, c, g, or t
<220>
<221> modified_base
<222> (20)..(20)
<223> a, c, g, or u
<400> 209
nnnnnnnnnn nnnnnnnnnn 20
<210> 210
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> d20
<220>
<221> modified_base
<222> (1)..(19)
<223> a, c, g, or u
<220>
<221> modified_base
<222> (20)..(20)
<223> a, c, g, or t
<400> 210
nnnnnnnnnn nnnnnnnnnn 20
<210> 211
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> BM.t12_cr
<400> 211
agugggggug aauucagugu 20
<210> 212
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.t12_d1
<400> 212
agugggggug aauucagugu 20
<210> 213
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.t12_d2
<400> 213
agugggggug aauucagugu 20
<210> 214
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.t12_d3
<400> 214
agtgggggug aauucagugu 20
<210> 215
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.t12_d4
<400> 215
agugggggug aauucagugu 20
<210> 216
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.t12_d5
<400> 216
agugggggug aauucagugu 20
<210> 217
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.t12_d6
<400> 217
agugggggug aauucagugu 20
<210> 218
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.t12_d7
<400> 218
agugggggug aauucagugu 20
<210> 219
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.t12_d8
<400> 219
agugggggug aauucagugu 20
<210> 220
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.t12_d9
<400> 220
agugggggtg aauucagugu 20
<210> 221
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.t12_d10
<400> 221
agugggggug aauucagugu 20
<210> 222
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.t12_d11
<400> 222
agugggggug aauucagugu 20
<210> 223
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.t12_d12
<400> 223
agugggggug aauucagugu 20
<210> 224
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.t12_d13
<400> 224
agugggggug aatucagugu 20
<210> 225
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.t12_d14
<400> 225
agugggggug aautcagugu 20
<210> 226
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.t12_d15
<400> 226
agugggggug aauucagugu 20
<210> 227
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.t12_d16
<400> 227
agugggggug aauucagugu 20
<210> 228
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.t12_d17
<400> 228
agugggggug aauucagugu 20
<210> 229
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.t12_d18
<400> 229
agugggggug aauucagtgu 20
<210> 230
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.t12_d19
<400> 230
agugggggug aauucagugu 20
<210> 231
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.t12_d20
<400> 231
agugggggug aauucagugt 20
<210> 232
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> TR.t12_cr
<400> 232
gagucucuca gcugguacac 20
<210> 233
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TR.t12_d1
<400> 233
gagucucuca gcugguacac 20
<210> 234
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TR.t12_d2
<400> 234
gagucucuca gcugguacac 20
<210> 235
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TR.t12_d3
<400> 235
gagucucuca gcugguacac 20
<210> 236
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TR.t12_d4
<400> 236
gagtcucuca gcugguacac 20
<210> 237
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TR.t12_d5
<400> 237
gagucucuca gcugguacac 20
<210> 238
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TR.t12_d6
<400> 238
gaguctcuca gcugguacac 20
<210> 239
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TR.t12_d7
<400> 239
gagucucuca gcugguacac 20
<210> 240
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TR.t12_d8
<400> 240
gagucuctca gcugguacac 20
<210> 241
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TR.t12_d9
<400> 241
gagucucuca gcugguacac 20
<210> 242
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TR.t12_d10
<400> 242
gagucucuca gcugguacac 20
<210> 243
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TR.t12_d11
<400> 243
gagucucuca gcugguacac 20
<210> 244
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TR.t12_d12
<400> 244
gagucucuca gcugguacac 20
<210> 245
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TR.t12_d13
<400> 245
gagucucuca gctgguacac 20
<210> 246
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TR.t12_d14
<400> 246
gagucucuca gcugguacac 20
<210> 247
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TR.t12_d15
<400> 247
gagucucuca gcugguacac 20
<210> 248
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TR.t12_d16
<400> 248
gagucucuca gcuggtacac 20
<210> 249
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TR.t12_d17
<400> 249
gagucucuca gcugguacac 20
<210> 250
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TR.t12_d18
<400> 250
gagucucuca gcugguacac 20
<210> 251
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TR.t12_d19
<400> 251
gagucucuca gcugguacac 20
<210> 252
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TR.t12_d20
<400> 252
gagucucuca gcugguacac 20
<210> 253
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> DMT.t3_cr
<400> 253
cugauggucc augucuguua 20
<210> 254
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_d1
<400> 254
cugauggucc augucuguua 20
<210> 255
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_d2
<400> 255
ctgauggucc augucuguua 20
<210> 256
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_d3
<400> 256
cugauggucc augucuguua 20
<210> 257
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_d4
<400> 257
cugauggucc augucuguua 20
<210> 258
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_d5
<400> 258
cugatggucc augucuguua 20
<210> 259
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_d6
<400> 259
cugauggucc augucuguua 20
<210> 260
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_d7
<400> 260
cugauggucc augucuguua 20
<210> 261
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_d8
<400> 261
cugauggtcc augucuguua 20
<210> 262
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_d9
<400> 262
cugauggucc augucuguua 20
<210> 263
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_d10
<400> 263
cugauggucc augucuguua 20
<210> 264
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_d11
<400> 264
cugauggucc augucuguua 20
<210> 265
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_d12
<400> 265
cugauggucc atgucuguua 20
<210> 266
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_d13
<400> 266
cugauggucc augucuguua 20
<210> 267
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_d14
<400> 267
cugauggucc augtcuguua 20
<210> 268
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_d15
<400> 268
cugauggucc augucuguua 20
<210> 269
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_d16
<400> 269
cugauggucc auguctguua 20
<210> 270
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_d17
<400> 270
cugauggucc augucuguua 20
<210> 271
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_d18
<400> 271
cugauggucc augucugtua 20
<210> 272
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_d19
<400> 272
cugauggucc augucuguta 20
<210> 273
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_d20
<400> 273
cugauggucc augucuguua 20
<210> 274
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> BM_tgt1_act.3'_crRNA
<400> 274
auauaagugg aggcgucgcg 20
<210> 275
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM_tgt1_act.3'_d.1
<400> 275
auauaagugg aggcgucgcg 20
<210> 276
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM_tgt1_act.3'_d.2
<400> 276
atauaagugg aggcgucgcg 20
<210> 277
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM_tgt1_act.3'_d.3
<400> 277
auauaagugg aggcgucgcg 20
<210> 278
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM_tgt1_act.3'_d.4
<400> 278
auataagugg aggcgucgcg 20
<210> 279
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM_tgt1_act.3'_d.5
<400> 279
auauaagugg aggcgucgcg 20
<210> 280
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM_tgt1_act.3'_d.6
<400> 280
auauaagugg aggcgucgcg 20
<210> 281
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM_tgt1_act.3'_d.7
<400> 281
auauaagugg aggcgucgcg 20
<210> 282
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM_tgt1_act.3'_d.8
<400> 282
auauaagtgg aggcgucgcg 20
<210> 283
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM_tgt1_act.3'_d.9
<400> 283
auauaagugg aggcgucgcg 20
<210> 284
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM_tgt1_act.3'_d.10
<400> 284
auauaagugg aggcgucgcg 20
<210> 285
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM_tgt1_act.3'_d.11
<400> 285
auauaagugg aggcgucgcg 20
<210> 286
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM_tgt1_act.3'_d.12
<400> 286
auauaagugg aggcgucgcg 20
<210> 287
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM_tgt1_act.3'_d.13
<400> 287
auauaagugg aggcgucgcg 20
<210> 288
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM_tgt1_act.3'_d.14
<400> 288
auauaagugg aggcgucgcg 20
<210> 289
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM_tgt1_act.3'_d.15
<400> 289
auauaagugg aggcgucgcg 20
<210> 290
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM_tgt1_act.3'_d.16
<400> 290
auauaagugg aggcgtcgcg 20
<210> 291
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM_tgt1_act.3'_d.17
<400> 291
auauaagugg aggcgucgcg 20
<210> 292
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM_tgt1_act.3'_d.18
<400> 292
auauaagugg aggcgucgcg 20
<210> 293
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM_tgt1_act.3'_d.19
<400> 293
auauaagugg aggcgucgcg 20
<210> 294
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM_tgt1_act.3'_d.20
<400> 294
auauaagugg aggcgucgcg 20
<210> 295
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> BM.in_t12_act.3'_crRNA
<400> 295
ucuggaggcu cucaaggacu 20
<210> 296
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.in_t12_cr-3'_d.1
<400> 296
tcuggaggcu cucaaggacu 20
<210> 297
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.in_t12_cr-3'_d.2
<400> 297
ucuggaggcu cucaaggacu 20
<210> 298
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.in_t12_cr-3'_d.3
<400> 298
uctggaggcu cucaaggacu 20
<210> 299
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.in_t12_cr-3'_d.4
<400> 299
ucuggaggcu cucaaggacu 20
<210> 300
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.in_t12_cr-3'_d.5
<400> 300
ucuggaggcu cucaaggacu 20
<210> 301
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.in_t12_cr-3'_d.6
<400> 301
ucuggaggcu cucaaggacu 20
<210> 302
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.in_t12_cr-3'_d.7
<400> 302
ucuggaggcu cucaaggacu 20
<210> 303
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.in_t12_cr-3'_d.8
<400> 303
ucuggaggcu cucaaggacu 20
<210> 304
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.in_t12_cr-3'_d.9
<400> 304
ucuggaggcu cucaaggacu 20
<210> 305
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.in_t12_cr-3'_d.10
<400> 305
ucuggaggct cucaaggacu 20
<210> 306
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.in_t12_cr-3'_d.11
<400> 306
ucuggaggcu cucaaggacu 20
<210> 307
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.in_t12_cr-3'_d.12
<400> 307
ucuggaggcu ctcaaggacu 20
<210> 308
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.in_t12_cr-3'_d.13
<400> 308
ucuggaggcu cucaaggacu 20
<210> 309
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.in_t12_cr-3'_d.14
<400> 309
ucuggaggcu cucaaggacu 20
<210> 310
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.in_t12_cr-3'_d.15
<400> 310
ucuggaggcu cucaaggacu 20
<210> 311
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.in_t12_cr-3'_d.16
<400> 311
ucuggaggcu cucaaggacu 20
<210> 312
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.in_t12_cr-3'_d.17
<400> 312
ucuggaggcu cucaaggacu 20
<210> 313
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.in_t12_cr-3'_d.18
<400> 313
ucuggaggcu cucaaggacu 20
<210> 314
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.in_t12_cr-3'_d.19
<400> 314
ucuggaggcu cucaaggacu 20
<210> 315
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.in_t12_cr-3'_d.20
<400> 315
ucuggaggcu cucaaggact 20
<210> 316
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> TR.t12_cr
<400> 316
gagucucuca gcugguacac 20
<210> 317
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TR.t12_d20.19
<400> 317
gagucucuca gcugguacac 20
<210> 318
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TR.t12_d20.17
<400> 318
gagucucuca gcugguacac 20
<210> 319
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TR.t12_d19.15
<400> 319
gagucucuca gcugguacac 20
<210> 320
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TR.t12_d11.10
<400> 320
gagucucuca gcugguacac 20
<210> 321
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TR.t12_d20.11.1
<400> 321
gagucucuca gcugguacac 20
<210> 322
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TR.t12_d17.11
<400> 322
gagucucuca gcugguacac 20
<210> 323
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TR.t12_d2.1
<400> 323
gagucucuca gcugguacac 20
<210> 324
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TR.t12_d15.10.11
<400> 324
gagucucuca gcugguacac 20
<210> 325
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TR.t12_d20.17.10.1
<400> 325
gagucucuca gcugguacac 20
<210> 326
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TR.t12_d18.15.2
<400> 326
gagucucuca gcugguacac 20
<210> 327
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TR.t12_d19.15.11.10.3
<400> 327
gagucucuca gcugguacac 20
<210> 328
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TR.t12_d20.17.15.10.1
<400> 328
gagucucuca gcugguacac 20
<210> 329
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TR.t12_d_7
<400> 329
gagucucuca gcugguacac 20
<210> 330
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> BM_tgt1_act.3'_crRNA
<400> 330
auauaagugg aggcgucgcg 20
<210> 331
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.t12_d20.17
<400> 331
agugggggug aauucagugt 20
<210> 332
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.t12_d15.11
<400> 332
agugggggug aauucagugu 20
<210> 333
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.t12_d15.14
<400> 333
agugggggug aautcagugu 20
<210> 334
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.t12_d11.10
<400> 334
agugggggug aauucagugu 20
<210> 335
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.t12_d20.11.1
<400> 335
agugggggug aauucagugt 20
<210> 336
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.t12_d17.11
<400> 336
agugggggug aauucagugu 20
<210> 337
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.t12_d2.1
<400> 337
agugggggug aauucagugu 20
<210> 338
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.t12_d20.17.15.2
<400> 338
agugggggug aauucagugt 20
<210> 339
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.t12_d15.14.11.10
<400> 339
agugggggug aautcagugu 20
<210> 340
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.t12_d_6a
<400> 340
agugggggug aauucagugt 20
<210> 341
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.t12_d_8
<400> 341
agtgggggtg aautcagugu 20
<210> 342
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.t12_d_6b
<400> 342
agugggggug aatucagugu 20
<210> 343
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.t12_d16.9.1-3
<400> 343
agtgggggtg aauucagugu 20
<210> 344
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.t12_d_10
<400> 344
agugggggug aattcagtgt 20
<210> 345
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> BM_tgt1_cr-3'
<400> 345
auauaagugg aggcgucgcg 20
<210> 346
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM_tgt1_cr-3'_d.20.11.1
<400> 346
auauaagugg aggcgucgcg 20
<210> 347
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM_tgt1_cr-3'_d.19.14.8
<400> 347
auauaagtgg aggcgucgcg 20
<210> 348
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM_tgt1_cr-3'_d.19.17.14.8
<400> 348
auauaagtgg aggcgucgcg 20
<210> 349
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM_tgt1_cr-3'_d.12.11.8
<400> 349
auauaagugg aggcgucgcg 20
<210> 350
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM_tgt1_cr-3'_d.18.16.10
<400> 350
auauaagugg aggcgtcgcg 20
<210> 351
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM_tgt1_cr-3'_d.19.17.15.1
<400> 351
auauaagugg aggcgucgcg 20
<210> 352
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM_tgt1_cr-3'_d.19-16
<400> 352
auauaagugg aggcgtcgcg 20
<210> 353
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> BM.in_t12_cr-3'
<400> 353
ucuggaggcu cucaaggacu 20
<210> 354
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.in_t12_cr-3'_d.01.11.20
<400> 354
tcuggaggcu cucaaggact 20
<210> 355
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.in_t12_cr-3'_d.11.17.20
<400> 355
ucuggaggcu cucaaggact 20
<210> 356
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.in_t12_cr-3'_d.08.09.10.11
<400> 356
ucuggaggct cucaaggacu 20
<210> 357
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.in_t12_cr-3'_d.01.08.10.17
<400> 357
tcuggaggct cucaaggacu 20
<210> 358
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.in_t12_cr-3'_d.17.19.20
<400> 358
ucuggaggcu cucaaggact 20
<210> 359
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.in_t12_cr-3'_d.08.09.19.20
<400> 359
ucuggaggcu cucaaggact 20
<210> 360
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> BM.in_t12_cr-3'_d01.10.11.17.19.20
<400> 360
tcuggaggct cucaaggact 20
<210> 361
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> DNMt_crRNA
<400> 361
cugauggucc augucuguua 20
<210> 362
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_d1.20
<400> 362
cugauggucc augucuguua 20
<210> 363
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_d14.20
<400> 363
cugauggucc augtcuguua 20
<210> 364
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_d8.19
<400> 364
cugauggtcc augucuguta 20
<210> 365
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_d8.14
<400> 365
cugauggtcc augtcuguua 20
<210> 366
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_d1.8.20
<400> 366
cugauggtcc augucuguua 20
<210> 367
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_d8-10
<400> 367
cugauggtcc augucuguua 20
<210> 368
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_d8.9.15
<400> 368
cugauggtcc augucuguua 20
<210> 369
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_d1.8.19
<400> 369
cugauggtcc augucuguta 20
<210> 370
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> RPL32_on
<400> 370
tttggggtga tcagacccaa cagc 24
<210> 371
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> RPL_32-off.1
<400> 371
tttggggtga tcagacccaa cacc 24
<210> 372
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> RPL_32-off.2
<400> 372
tttggggtga tcagacccaa cacc 24
<210> 373
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> DNMT1_on
<400> 373
tttcctgatg gtccatgtct gtta 24
<210> 374
<211> 24
<212> DNA
<213> Homo sapiens
<220>
<223> DNMT1_off-1
<400> 374
tttcctgatg gtccatgtct gaat 24
<210> 375
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> RP_cr
<400> 375
gggugaucag acccaacagc 20
<210> 376
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> RP_cr_2_d.1
<400> 376
gggugaucag acccaacagc 20
<210> 377
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> RP_cr_2_d.8
<400> 377
gggugaucag acccaacagc 20
<210> 378
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> RP_cr_2_d.9
<400> 378
gggugaucag acccaacagc 20
<210> 379
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> RP_cr_2_d.10
<400> 379
gggugaucag acccaacagc 20
<210> 380
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> RP_cr_2_d.11
<400> 380
gggugaucag acccaacagc 20
<210> 381
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> RP_cr_2_d.12
<400> 381
gggugaucag acccaacagc 20
<210> 382
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> RP_cr_2_d.14
<400> 382
gggugaucag acccaacagc 20
<210> 383
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> RP_cr_2_d.15
<400> 383
gggugaucag acccaacagc 20
<210> 384
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> RP_cr_2_d.16
<400> 384
gggugaucag acccaacagc 20
<210> 385
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> RP_cr_2_d.17
<400> 385
gggugaucag acccaacagc 20
<210> 386
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> RP_cr_2_d.18
<400> 386
gggugaucag acccaacagc 20
<210> 387
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> RP_cr_2_d.19
<400> 387
gggugaucag acccaacagc 20
<210> 388
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> RP_cr_2_d.20
<400> 388
gggugaucag acccaacagc 20
<210> 389
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> RP_cr_3'_d.13
<400> 389
gggugaucag acccaacagc 20
<210> 390
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> RP_cr_3'_d11.12
<400> 390
gggugaucag acccaacagc 20
<210> 391
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> RP_cr_3'_d12.14
<400> 391
gggugaucag acccaacagc 20
<210> 392
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> RP_cr_3'_d14.15
<400> 392
gggugaucag acccaacagc 20
<210> 393
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> RP_cr_3'_d11.15
<400> 393
gggugaucag acccaacagc 20
<210> 394
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> RP_cr_3'_d11.12.14
<400> 394
gggugaucag acccaacagc 20
<210> 395
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> RP_cr_3'_d12.14.15
<400> 395
gggugaucag acccaacagc 20
<210> 396
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> RP_cr_3'_d11.14.15
<400> 396
gggugaucag acccaacagc 20
<210> 397
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> RP_cr_3'_d11.12.14.15
<400> 397
gggugaucag acccaacagc 20
<210> 398
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> RP_cr_3'_sp.20
<220>
<221> modified_base
<222> (20)..(20)
<223> an abasic site that lacks 2' - hydroxyl
<400> 398
gggugaucag acccaacagn t 21
<210> 399
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> RP_cr_3'_d14_sp20
<220>
<221> modified_base
<222> (20)..(20)
<223> an abasic site that lacks 2' - hydroxyl
<400> 399
gggugaucag acccaacagn t 21
<210> 400
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> RP_cr_3'_d15_sp20
<220>
<221> modified_base
<222> (20)..(20)
<223> an abasic site that lacks 2' - hydroxyl
<400> 400
gggugaucag acccaacagn t 21
<210> 401
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> RP_cr_3'_d12_sp20
<220>
<221> modified_base
<222> (20)..(20)
<223> an abasic site that lacks 2' - hydroxyl
<400> 401
gggugaucag acccaacagn t 21
<210> 402
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> RP_cr_3'_d11_sp20
<220>
<221> modified_base
<222> (20)..(20)
<223> an abasic site that lacks 2' - hydroxyl
<400> 402
gggugaucag acccaacagn t 21
<210> 403
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> RP_cr_3'_d14.15_sp20
<220>
<221> modified_base
<222> (20)..(20)
<223> an abasic site that lacks 2' - hydroxyl
<400> 403
gggugaucag acccaacagn t 21
<210> 404
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> DNMT1-crRNA
<400> 404
cugauggucc augucuguua 20
<210> 405
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DNMT1-d1.20
<400> 405
cugauggucc augucuguua 20
<210> 406
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DNMT1-d8.19
<400> 406
cugauggtcc augucuguta 20
<210> 407
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DNMT1-d9
<400> 407
cugauggucc augucuguua 20
<210> 408
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DNMT1-d8.14
<400> 408
cugauggtcc augtcuguua 20
<210> 409
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DNMT1-d10.17
<400> 409
cugauggucc augucuguua 20
<210> 410
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DNMT1-d1.8.20
<400> 410
cugauggtcc augucuguua 20
<210> 411
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DNMT1-d1.8.19
<400> 411
cugauggtcc augucuguta 20
<210> 412
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DNMT1-d8.9.15
<400> 412
cugauggtcc augucuguua 20
<210> 413
<211> 3147
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polynucleotide
<220>
<223> anti-BCMA CAR construct
<400> 413
cataaacctc ccattctgct aatgcccagc ctaagttggg gagaccactc cagattccaa 60
gatgtacagt ttgctttgct gggccttttt cccatgcctg cctttactct gccagagtta 120
tattgctggg gttttgaaga agatcctatt aaataaaaga ataagcagta ttattaagta 180
gccctgcatt tcaggtttcc ttgagtggca ggccaggcct ggccgtgaac gttcactgaa 240
atcatggcct cttggccaag attgatagct tgtgcctgtc cctgagtccc agtccatcac 300
gagcagctgg tttctaagat gctatttccc gtataaagca tgagaccgtg acttgccagc 360
cccacagagc cccgcccttg tccatcactg gcatctggac tccagcctgg gttggggcaa 420
agagggaaat gagatcatgt cctaaccctg atcctcttgt cccacagata tccagaaccc 480
tgaccctgcc gtgtaccagc ttaattaatt agtccataga gcccaccgca tccccagcat 540
gcctgctatt gtcttcccaa tcctccccct tgctgtcctg ccccacccca ccccccagaa 600
tagaatgaca cctactcaga caatgcgatg caatttcctc attttattag gaaaggacag 660
tgggagtggc accttccagg gtcaaggaag gcacggggga ggggcaaaca acagatggct 720
ggcaactaga aggcacagtt attacctcgg tggcagggcc tgcatatgga gggcatcgta 780
tgtatctttc gtggccgtac tcaggccttg gtagagaccg tcgtggcctt tccctctgcg 840
tctctctccc ttcatcccga tctcgctgta agcctccgcc atcttatctt tctgcagttc 900
attataaagc ccctcttgcg ggttttttct cctcggtttt cctcccatct ctgggtccct 960
ccctcggcgc ttatccaata catcatattc ctctcgcctc cccaggttaa gctcattgta 1020
aagttggttt tgcccttgct gataggccgg agcgtcagca gaccggctaa atttcactct 1080
cagttcgcaa ccaccctcct cttcttcggg gaatcggcag ctacatccat cctcctcttg 1140
cgtagtttgg accgggcgca taaatggttg cttaaaaata tacagcaact tttttcttcc 1200
tcttttgcag taaagagtaa taacgaggga cagcagcaag accccgcacg ttccggcaag 1260
cggtgcccag atgtagatgt cgcaagcaaa gtccagcccg cgagtatgaa ccgcgcctcc 1320
cgctgcgggg cgacaagcct ccggccgaag agagagtggc tgggaagcga tagtgggtgc 1380
aggcgtgggt ggccgagggg caggcgtggt tgtggcagcg gctttaattt ccactttggt 1440
gccgccgcca aaggtcggcg gaaagctatg gccgttctgg caataataca ccgcaaaatc 1500
ttccggttcc aggctgctaa tggtcagggt aaaatcggtg ccgctgccgc tgccgctaaa 1560
gcgcgccgga atgccggtaa tgctctggct cgcataataa atcagcaggc gcggcgcctg 1620
gcccggtttc tgctgatacc aatgcagata atcgctaatg ctctggctcg cgcggcagct 1680
cagggtcgcg cgttcgcccg ggctcaggct cagggtcgcc gggctctggg tcagcacaat 1740
ttcgctgccg ccgccgccgc tgccgccgcc gccgctgccg ccgccgccgc tgctcacggt 1800
caccagggtg ccctggcccc acacatcaaa atagccatca tagttatagc gcgcgcaata 1860
atacaccgcg gtatcttcgc tgcgcaggct gctcagttcc atatacgcgg tgctggtgct 1920
tttatccgcg gtaatggtca cgcggccttt aaatttttcg ttatatttgg tcgcatcgtt 1980
atacggaata atatagccca tccattccag gccctggccc ggcgcctggc gcacccaatg 2040
catcacatag ctggtaaagg tatagccgct cgctttgcag ctcactttca cgctgctgcc 2100
cggttttttc acttccgcgc cgctctgcac cagctgcacc tgcggtcttg ccgcgtggag 2160
caggagtgcc aatggaagca gaagtgcggt cacgggcaac gccatggtgg ctatgacgcg 2220
tcgcgccgag tgaggggttg tgggctcttt tattgagctc ggggagcaga agcgcgcgaa 2280
cagaagcgag aagcgaactg attggttagt tcaaataagg cacagggtca tttcaggtcc 2340
ttggggcacc ctggaaacat ctgatggttc tctagaaact gctgagggcg ggaccgcatc 2400
tggggaccat ctgttcttgg ccctgagccg gggcaggaac tgcttaccac agatatcctg 2460
tttggcccat attctgctgt tccaactgtt cttggccctg agccggggca ggaactgctt 2520
accacagata tcctgtttgg cccatattct gctgtctctc tgttcctaac cttgatccta 2580
gcttgccaaa cctacaggtg gggtctttca ttcccccctt tttctggaga ctaaataaag 2640
tttaaactga gagactctaa atccagtgac aagtctgtct gcctattcac cgattttgat 2700
tctcaaacaa atgtgtcaca aagtaaggat tctgatgtgt atatcacaga caaaactgtg 2760
ctagacatga ggtctatgga cttcaagagc aacagtgctg tggcctggag caacaaatct 2820
gactttgcat gtgcaaacgc cttcaacaac agcattattc cagaagacac cttcttcccc 2880
agcccaggta agggcagctt tggtgccttc gcaggctgtt tccttgcttc aggaatggcc 2940
aggttctgcc cagagctctg gtcaatgatg tctaaaactc ctctgattgg tggtctcggc 3000
cttatccatt gccaccaaaa ccctcttttt actaagaaac agtgagcctt gttctggcag 3060
tccagagaat gacacgggaa aaaagcagat gaagagaagg tggcaggaga gggcacgtgg 3120
cccagcctca gtctctccaa ctgagtt 3147
<210> 414
<211> 3409
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polynucleotide
<220>
<223> B2M-HLA-e construct
<400> 414
tgagtgctga gagggcatca gaagtccttg agagcctcca gagaaaggct cttaaaaatg 60
cagcgcaatc tccagtgaca gaagatactg ctagaaatct gctagaaaaa aaacaaaaaa 120
ggcatgtata gaggaattat gagggaaaga taccaagtca cggtttattc ttcaaaatgg 180
aggtggcttg ttgggaaggt ggaagctcat ttggccagag tggaaatgga attgggagaa 240
atcgatgacc aaatgtaaac acttggtgcc tgatatagct tgacaccaag ttagccccaa 300
gtgaaatacc ctggcaatat taatgtgtct tttcccgata ttcctcaggt actccaaaga 360
ttcaggttta ctcacgtcat ccagcagaga atggaaagtc aaatttcctg aattgctatg 420
tgtctgggtt tcatccatcc gacattgaag ttgacttact gaagaatgga gagagaattg 480
aaaaagtgga gcattcagac ttgtctttca gcaaggactg gtctttctat ctcttgtact 540
acactgaatt tttaattaat tagtccatag agcccaccgc atccccagca tgcctgctat 600
tgtcttccca atcctccccc ttgctgtcct gccccacccc accccccaga atagaatgac 660
acctactcag acaatgcgat gcaatttcct cattttatta ggaaaggaca gtgggagtgg 720
caccttccag ggtcaaggaa ggcacggggg aggggcaaac aacagatggc tggcaactag 780
aaggcacagt tattacaagc tgtgagactc agacccctgg gcactgtcgc tccactcagc 840
cttagagtag ctccctcctt ttccacctga gctcttcttc ctccatatca cagcagcaac 900
cacagctcca gagaccacag atccaaggag aaccaggcca gcaatgatgc ccacgatggg 960
gatggtgggc tgggaagccg gcttccatct cagggtgacg ggctcgggta gcccctcatg 1020
ctgcacatgg cacgtgtatc tctgctcctc tccagaaggc accaccacag ctgcccactt 1080
ctggaaggtt ccatcccctg caggcctggt ctccacgagc tccgtgtcct gggtatggcc 1140
ctccccatcc tgctgccagg tcagtgtgat ctccgcaggg tagaagccca gggcccagca 1200
cctcagggtg gcctcatggt cagagatggg gtggtgagtc acgtgtgtct ttgggggctc 1260
caggtgaagc agcgtctcct tccccttctc caggtatttg tggagccact ccacgcatgt 1320
gtcttccagg taggctctct ggtgctccgc ctcagaagca tcatttgact tttgctcgga 1380
gatctgagcc gccgtgtcca ccgcggtcca ggagcgcagg tcctcattca gggtgagata 1440
atccttgccg tcgtaggcga actgttcata cccgcggagg aagcgcccgt cgggccccag 1500
ctcgcagcca tgcatccact gcagggtgtg agacccggcc tcgctctgat tgtagtagcc 1560
gcgcagcgtc cgcagattca ctcggaaaat ctgtgcggtg tccctggcgc tccgtgtctc 1620
ccggtcccaa tactctgacc cctcctgctc catccacggc gcccgcggca ccatcctcgg 1680
actcgcggcg tcgttgtcga agcgcacgaa ctgggtgtcg tccacgtagc ccacagagat 1740
gaagcggggc tccccgcggc cgggccggga cacggaagtg tggaaatact tcaaggagtg 1800
ggatccagac cctccgccac cagatccccc tcctccagaa ccgcctccgc cagaccctcc 1860
gccacccatg tctcgatccc atttcacgat cttgggctgt gacaaagtca catggttcac 1920
acggcaggca tactcatctt tctccgtagg tgtaaactct gtatagtaca agagatagaa 1980
agaccagtcc ttgctgaaag acaagtctga atgctccact ttttcaattc tctctccatt 2040
cttcagtaag tcaacttcaa tgtcggatgg atgaaaccca gacacatagc aattcaggaa 2100
atttgacttt ccattctctg ctggatgacg tgagtaaacc tgaatctttg gagtacgctg 2160
gatggagccg ccgcctccgc taccgcctcc tccgctccca cctccaccca gaaagagggt 2220
ccgtggtgcc attacagcct ccaggccaga aagagagagt agcgcgagca cagctaaggc 2280
cacggagcga gacatggtgg ctctagagta ggcgccggtc acagcttgga tctgtaacgg 2340
cgcagaacag aaaacgaaac aaagacgtag agttgagcaa gcagggtcag gcaaagcgtg 2400
gagagccggc tgagtctagg taggctccaa gggagcgccg gacaaaggcc cggtctcgac 2460
ctgagcttta aacttaccta gacggcggac gcagttcagg aggcaccaca ggcgggaggc 2520
ggcagaacgc gactcaaccg gcgtggatgg cggcctcagg tagggcggcg ggcgcgtgaa 2580
ggagagatgc gagcccctcg aagcttcagc tgtgttctgg cggcaaaccc gttgcgaaaa 2640
agaacgttca cggcgactac tgcacttata tacggttctc ccccaccctc gggaaaaagg 2700
cggagccagt acacgacatc actttcccag tttaccccgc gccaccttct ctaggcaccc 2760
gttcaattgc cgacccctcc ccccaacttc tcggggactg tgggcgatgt gcgctctgcc 2820
cactgacggg caccggagcg atcgcagatc cttgtcgacc acccccactg aaaaagatga 2880
gtatgcctgc cgtgtgaacc atgtgacttt gtcacagccc aagatagtta agtggggtaa 2940
gtcttacatt cttttgtaag ctgctgaaag ttgtgtatga gtagtcatat cataaagctg 3000
ctttgatata aaaaaggtct atggccatac taccctgaat gagtcccatc ccatctgata 3060
taaacaatct gcatattggg attgtcaggg aatgttctta aagatcagat tagtggcacc 3120
tgctgagata ctgatgcaca gcatggtttc tgaaccagta gtttccctgc agttgagcag 3180
ggagcagcag cagcacttgc acaaatacat atacactctt aacacttctt acctactggc 3240
ttcctctagc ttttgtggca gcttcaggta tatttagcac tgaacgaaca tctcaagaag 3300
gtataggcct ttgtttgtaa gtcctgctgt cctagcatcc tataatcctg gacttctcca 3360
gtactttctg gctggattgg tatctgaggc tagtaggaag ggcttgttc 3409
<210> 415
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TRAC_cr3'_tgt.12-d20.11.1
<400> 415
taauuucuac ucutguagau gagucucuca gcugguacac 40
<210> 416
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> B2M_cr3'_tgt.12-d20.11.1
<400> 416
taauuucuac ucutguagau agugggggug aauucagugt 40
<210> 417
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> crRNA
<400> 417
uaauuucuac ucuuguagau 20
<210> 418
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> CRISPR-d1
<400> 418
taauuucuac ucuuguagau 20
<210> 419
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> CRISPR-d2
<400> 419
uaauuucuac ucuuguagau 20
<210> 420
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> CRISPR-d3
<400> 420
uaauuucuac ucuuguagau 20
<210> 421
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> CRISPR-d4
<400> 421
uaatuucuac ucuuguagau 20
<210> 422
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> CRISPR-d5
<400> 422
uaautucuac ucuuguagau 20
<210> 423
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> CRISPR-d6
<400> 423
uaauutcuac ucuuguagau 20
<210> 424
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> CRISPR-d7
<400> 424
uaauuucuac ucuuguagau 20
<210> 425
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> CRISPR-d8
<400> 425
uaauuuctac ucuuguagau 20
<210> 426
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> CRISPR-d9
<400> 426
uaauuucuac ucuuguagau 20
<210> 427
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> CRISPR-d10
<400> 427
uaauuucuac ucuuguagau 20
<210> 428
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> CRISPR-d11
<400> 428
uaauuucuac tcuuguagau 20
<210> 429
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> CRISPR-d12
<400> 429
uaauuucuac ucuuguagau 20
<210> 430
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> CRISPR-d13
<400> 430
uaauuucuac uctuguagau 20
<210> 431
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> CRISPR-d14
<400> 431
uaauuucuac ucutguagau 20
<210> 432
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> CRISPR-d15
<400> 432
uaauuucuac ucuuguagau 20
<210> 433
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> CRISPR-d16
<400> 433
uaauuucuac ucuugtagau 20
<210> 434
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> CRISPR-d17
<400> 434
uaauuucuac ucuuguagau 20
<210> 435
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> CRISPR-d18
<400> 435
uaauuucuac ucuuguagau 20
<210> 436
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> CRISPR-d19
<400> 436
uaauuucuac ucuuguagau 20
<210> 437
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> CRISPR-d20
<400> 437
uaauuucuac ucuuguagat 20
<210> 438
<211> 40
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> DMT.t3_crRNA
<400> 438
uaauuucuac ucuuguagau cugauggucc augucuguua 40
<210> 439
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_cr-d1
<400> 439
taauuucuac ucuuguagau cugauggucc augucuguua 40
<210> 440
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_cr-d2
<400> 440
uaauuucuac ucuuguagau cugauggucc augucuguua 40
<210> 441
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_cr-d3
<400> 441
uaauuucuac ucuuguagau cugauggucc augucuguua 40
<210> 442
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_cr-d4
<400> 442
uaatuucuac ucuuguagau cugauggucc augucuguua 40
<210> 443
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_cr-d5
<400> 443
uaautucuac ucuuguagau cugauggucc augucuguua 40
<210> 444
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_cr-d6
<400> 444
uaauutcuac ucuuguagau cugauggucc augucuguua 40
<210> 445
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_cr-d7
<400> 445
uaauuucuac ucuuguagau cugauggucc augucuguua 40
<210> 446
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_cr-d8
<400> 446
uaauuuctac ucuuguagau cugauggucc augucuguua 40
<210> 447
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_cr-d9
<400> 447
uaauuucuac ucuuguagau cugauggucc augucuguua 40
<210> 448
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_cr-d10
<400> 448
uaauuucuac ucuuguagau cugauggucc augucuguua 40
<210> 449
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_cr-d11
<400> 449
uaauuucuac tcuuguagau cugauggucc augucuguua 40
<210> 450
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_cr-d12
<400> 450
uaauuucuac ucuuguagau cugauggucc augucuguua 40
<210> 451
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_cr-d13
<400> 451
uaauuucuac uctuguagau cugauggucc augucuguua 40
<210> 452
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_cr-d14
<400> 452
uaauuucuac ucutguagau cugauggucc augucuguua 40
<210> 453
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_cr-d15
<400> 453
uaauuucuac ucuuguagau cugauggucc augucuguua 40
<210> 454
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_cr-d16
<400> 454
uaauuucuac ucuugtagau cugauggucc augucuguua 40
<210> 455
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_cr-d17
<400> 455
uaauuucuac ucuuguagau cugauggucc augucuguua 40
<210> 456
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_cr-d18
<400> 456
uaauuucuac ucuuguagau cugauggucc augucuguua 40
<210> 457
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_cr-d19
<400> 457
uaauuucuac ucuuguagau cugauggucc augucuguua 40
<210> 458
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> DMT.t3_cr-d20
<400> 458
uaauuucuac ucuuguagat cugauggucc augucuguua 40
<210> 459
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> CRISRP-3'
<400> 459
taauuucuac ucutguagau 20
<210> 460
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> CRISRP-2
<400> 460
taauuucuac ucuugtagau 20
<210> 461
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> CRISRP-6
<400> 461
taauuucuac uctugtagau 20
<210> 462
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> CRISRP-401
<400> 462
uaauuucuac ucutguagau 20
<210> 463
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> CRISRP-402
<400> 463
taauuucuac ucutgtagau 20
<210> 464
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> CRISRP-403
<400> 464
uaauuucuac ucutguagau 20
<210> 465
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> CRISRP-404
<400> 465
taauuucuac ucutguagau 20
<210> 466
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TR_cr_tgt.12-d20.11.1
<400> 466
uaauuucuac ucuuguagau gagucucuca gcugguacac 40
<210> 467
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TR_cr3'_tgt.12-d20.11.1
<400> 467
taauuucuac ucutguagau gagucucuca gcugguacac 40
<210> 468
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TR_cr-2_tgt.12-d20.11.1
<400> 468
taauuucuac ucuugtagau gagucucuca gcugguacac 40
<210> 469
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TR_cr-6_tgt.12-d20.11.1
<400> 469
taauuucuac uctugtagau gagucucuca gcugguacac 40
<210> 470
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TR_cr-401_tgt.12-d20.11.1
<400> 470
uaauuucuac ucutguagau gagucucuca gcugguacac 40
<210> 471
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TR_cr-403_tgt.12-d20.11.1
<400> 471
taauuucuac ucutgtagau gagucucuca gcugguacac 40
<210> 472
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TR_cr-404_tgt.12-d20.11.1
<400> 472
uaauuucuac ucutguagau gagucucuca gcugguacac 40
<210> 473
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TR_cr-405_tgt.12-d20.11.1
<400> 473
taauuucuac ucutguagau gagucucuca gcugguacac 40
<210> 474
<211> 117
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<223> BCMA scFv chain 1
<400> 474
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Val Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Tyr Ile Ile Pro Tyr Asn Asp Ala Thr Lys Tyr Asn Glu Lys Phe
50 55 60
Lys Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Tyr Asn Tyr Asp Gly Tyr Phe Asp Val Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser
115
<210> 475
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<223> BCMA scFv chain 2
<400> 475
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Ile Ser Asp Tyr
20 25 30
Leu His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Tyr Ala Ser Gln Ser Ile Thr Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Gln Asn Gly His Ser Phe Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 476
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<220>
<223> BCMA scFv linker
<400> 476
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
1 5 10 15
<210> 477
<211> 240
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<223> BCMA scFv
<400> 477
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Val Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Tyr Ile Ile Pro Tyr Asn Asp Ala Thr Lys Tyr Asn Glu Lys Phe
50 55 60
Lys Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Tyr Asn Tyr Asp Gly Tyr Phe Asp Val Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
115 120 125
Gly Gly Gly Gly Ser Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu
130 135 140
Ser Leu Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln
145 150 155 160
Ser Ile Ser Asp Tyr Leu His Trp Tyr Gln Gln Lys Pro Gly Gln Ala
165 170 175
Pro Arg Leu Leu Ile Tyr Tyr Ala Ser Gln Ser Ile Thr Gly Ile Pro
180 185 190
Ala Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile
195 200 205
Ser Ser Leu Glu Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Asn Gly
210 215 220
His Ser Phe Pro Pro Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
225 230 235 240
<210> 478
<211> 63
<212> DNA
<213> Homo sapiens
<220>
<223> CD8-alpha signal sequence
<400> 478
atggcgttgc ccgtgaccgc acttctgctt ccattggcac tcctgctcca cgcggcaaga 60
ccg 63
<210> 479
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<220>
<223> GS_SV40-NLS
<400> 479
Gly Ser Pro Lys Lys Lys Arg Lys Val
1 5
<210> 480
<211> 18
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<220>
<223> GS_SV40-NLS_GS_SV40-NLS
<400> 480
Gly Ser Pro Lys Lys Lys Arg Lys Val Gly Ser Pro Lys Lys Lys Arg
1 5 10 15
Lys Val
<210> 481
<211> 18
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<220>
<223> GS_NPL-NLS
<400> 481
Gly Ser Lys Arg Pro Ala Ala Thr Lys Lys Ala Gly Gln Ala Lys Lys
1 5 10 15
Lys Lys
<210> 482
<211> 36
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<223> GS_NPL-NLS_GS_NPL-NLS
<400> 482
Gly Ser Lys Arg Pro Ala Ala Thr Lys Lys Ala Gly Gln Ala Lys Lys
1 5 10 15
Lys Lys Gly Ser Lys Arg Pro Ala Ala Thr Lys Lys Ala Gly Gln Ala
20 25 30
Lys Lys Lys Lys
35
<210> 483
<211> 27
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<220>
<223> GS_SV40-NLS_GS_NPL-NLS
<400> 483
Gly Ser Pro Lys Lys Lys Arg Lys Val Gly Ser Lys Arg Pro Ala Ala
1 5 10 15
Thr Lys Lys Ala Gly Gln Ala Lys Lys Lys Lys
20 25
<210> 484
<211> 27
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<220>
<223> GS_NPL-NLS_GS_SV40-NLS
<400> 484
Gly Ser Lys Arg Pro Ala Ala Thr Lys Lys Ala Gly Gln Ala Lys Lys
1 5 10 15
Lys Lys Gly Ser Pro Lys Lys Lys Arg Lys Val
20 25
<210> 485
<211> 35
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<223> (GGGGS)2_SV40-NLS_GS_NPL-NLS
<400> 485
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Pro Lys Lys Lys Arg Lys
1 5 10 15
Val Gly Ser Lys Arg Pro Ala Ala Thr Lys Lys Ala Gly Gln Ala Lys
20 25 30
Lys Lys Lys
35
<210> 486
<211> 35
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<223> (GGGGS)2_NPL-NLS_GS_SV40-NLS
<400> 486
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Lys Arg Pro Ala Ala Thr
1 5 10 15
Lys Lys Ala Gly Gln Ala Lys Lys Lys Lys Gly Ser Pro Lys Lys Lys
20 25 30
Arg Lys Val
35
<210> 487
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<220>
<223> (GGGGS)2_SV40-NLS
<400> 487
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Pro Lys Lys Lys Arg Lys
1 5 10 15
Val
<210> 488
<211> 26
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<220>
<223> (GGGGS)2_SV40-NLS_GS_SV40-NLS
<400> 488
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Pro Lys Lys Lys Arg Lys
1 5 10 15
Val Gly Ser Pro Lys Lys Lys Arg Lys Val
20 25
<210> 489
<211> 26
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<220>
<223> (GGGGS)2_NPL-NLS
<400> 489
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Lys Arg Pro Ala Ala Thr
1 5 10 15
Lys Lys Ala Gly Gln Ala Lys Lys Lys Lys
20 25
<210> 490
<211> 44
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<223> (GGGGS)2_NPL-NLS_GS_NPL-NLS
<400> 490
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Lys Arg Pro Ala Ala Thr
1 5 10 15
Lys Lys Ala Gly Gln Ala Lys Lys Lys Lys Gly Ser Lys Arg Pro Ala
20 25 30
Ala Thr Lys Lys Ala Gly Gln Ala Lys Lys Lys Lys
35 40
<210> 491
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> TRAC-tgt25
<400> 491
cagauacgaa ccuaaacuuu 20
<210> 492
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> TRAC-tgt28
<400> 492
aggaggagga uucggaaccc 20
<210> 493
<211> 21
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<220>
<223> 53BP1
<400> 493
Gly Lys Arg Lys Leu Ile Thr Ser Glu Glu Glu Arg Ser Pro Ala Lys
1 5 10 15
Arg Gly Arg Lys Ser
20
<210> 494
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<220>
<223> VACM-1/CUL5
<400> 494
Pro Lys Leu Lys Arg Gln
1 5
<210> 495
<211> 4
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<220>
<223> CXCR4
<400> 495
Arg Pro Arg Lys
1
<210> 496
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<220>
<223> VP1
<400> 496
Arg Arg Ala Arg Arg Pro Arg Gly
1 5
<210> 497
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<220>
<223> ING4
<400> 497
Lys Gly Lys Lys Gly Arg Thr Gln Lys Glu Lys Lys Ala Ala Arg Ala
1 5 10 15
Arg Ser Lys Gly Lys Asn
20
<210> 498
<211> 28
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<220>
<223> IER5
<400> 498
Arg Lys Arg Cys Ala Ala Gly Val Gly Gly Gly Pro Ala Gly Cys Pro
1 5 10 15
Ala Pro Gly Ser Thr Pro Leu Lys Lys Pro Arg Arg
20 25
<210> 499
<211> 36
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<223> ERK5
<400> 499
Arg Lys Pro Val Thr Ala Gln Glu Arg Gln Arg Glu Arg Glu Glu Lys
1 5 10 15
Arg Arg Arg Arg Gln Glu Arg Ala Lys Glu Arg Glu Lys Arg Arg Gln
20 25 30
Glu Arg Glu Arg
35
<210> 500
<211> 27
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<220>
<223> UL79
<400> 500
Thr Leu Leu Leu Arg Glu Thr Met Asn Asn Leu Gly Val Ser Asp His
1 5 10 15
Ala Val Leu Ser Arg Lys Thr Pro Gln Pro Tyr
20 25
<210> 501
<211> 18
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<220>
<223> EWS
<400> 501
Pro Gly Lys Met Asp Lys Gly Glu His Arg Gln Glu Arg Arg Asp Arg
1 5 10 15
Pro Tyr
<210> 502
<211> 27
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<220>
<223> Hrp1
<400> 502
Arg Ser Gly Gly Asn His Arg Arg Asn Gly Arg Gly Gly Arg Gly Gly
1 5 10 15
Tyr Asn Arg Arg Asn Asn Gly Tyr His Pro Tyr
20 25
<210> 503
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<220>
<223> cMyc (1)
<400> 503
Pro Ala Ala Lys Arg Cys Lys Leu Asp
1 5
<210> 504
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<220>
<223> cMyc (2)
<400> 504
Arg Gln Arg Arg Asn Glu Leu Lys Arg Ser Pro
1 5 10
<210> 505
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<220>
<223> mouse c-able IV
<400> 505
Ser Ala Leu Ile Lys Lys Lys Lys Lys Asn Ala Pro
1 5 10
<210> 506
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<220>
<223> Matalpha2
<400> 506
Lys Ile Pro Ile Lys
1 5
<210> 507
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<220>
<223> MINIYO
<400> 507
Lys Ser Gly Ser Arg Lys
1 5
<210> 508
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TRAC-tgt25 chRDNA
<400> 508
taauuucuac ucutguagau cagauacgaa ccuaaacuut 40
<210> 509
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> CISH-tgt3 chRDNA
<400> 509
taauuucuac ucutguagau gguguacagc aguggcuggu 40
<210> 510
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> CBLB-tgt3 chRDNA
<400> 510
taauuucuac ucutguagau ccugauacau aucagcauuu 40
<210> 511
<211> 40
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> TRAC-tgt12 crRNA
<400> 511
uaauuucuac ucuuguagau gagucucuca gcugguacac 40
<210> 512
<211> 40
<212> RNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> TRAC-tgt12 ps-r1.2_s19.20
<220>
<221> misc_feature
<222> (1)..(3)
<223> Phosphorothioate linkage
<220>
<221> misc_feature
<222> (38)..(40)
<223> Phosphorothioate linkage
<400> 512
uaauuucuac ucuuguagau gagucucuca gcugguacac 40
<210> 513
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TRAC-tgt12 ps-r2_r-d1
<220>
<221> misc_feature
<222> (1)..(3)
<223> Phosphorothioate linkage
<220>
<221> misc_feature
<222> (38)..(40)
<223> Phosphorothioate linkage
<400> 513
taauuucuac ucuuguagau gagucucuca gcugguacac 40
<210> 514
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TRAC-tgt12 ps-r1.2_r-d7
<220>
<221> misc_feature
<222> (1)..(3)
<223> Phosphorothioate linkage
<220>
<221> misc_feature
<222> (7)..(8)
<223> Phosphorothioate linkage
<220>
<221> misc_feature
<222> (38)..(40)
<223> Phosphorothioate linkage
<400> 514
uaauuucuac ucuuguagau gagucucuca gcugguacac 40
<210> 515
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TRAC-tgt12 ps-r1.2_r-d10
<220>
<221> misc_feature
<222> (1)..(3)
<223> Phosphorothioate linkage
<220>
<221> misc_feature
<222> (10)..(11)
<223> Phosphorothioate linkage
<220>
<221> misc_feature
<222> (38)..(40)
<223> Phosphorothioate linkage
<400> 515
uaauuucuac ucuuguagau gagucucuca gcugguacac 40
<210> 516
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TRAC-tgt12 ps-r1.2_r-d12
<220>
<221> misc_feature
<222> (1)..(3)
<223> Phosphorothioate linkage
<220>
<221> misc_feature
<222> (12)..(13)
<223> Phosphorothioate linkage
<220>
<221> misc_feature
<222> (38)..(40)
<223> Phosphorothioate linkage
<400> 516
uaauuucuac ucuuguagau gagucucuca gcugguacac 40
<210> 517
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> TRAC-tgt12 ps-r1.2_r-d14
<220>
<221> misc_feature
<222> (1)..(3)
<223> Phosphorothioate linkage
<220>
<221> misc_feature
<222> (14)..(15)
<223> Phosphorothioate linkage
<220>
<221> misc_feature
<222> (38)..(40)
<223> Phosphorothioate linkage
<400> 517
uaauuucuac ucutguagau gagucucuca gcugguacac 40
<210> 518
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> CSH-tgt3_3'_d1
<400> 518
taauuucuac ucutguagau gguguacagc aguggcuggu 40
<210> 519
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> CSH-tgt3_3'_d8
<400> 519
taauuucuac ucutguagau gguguacagc aguggcuggu 40
<210> 520
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> CSH-tgt3_3'_d9
<400> 520
taauuucuac ucutguagau gguguacagc aguggcuggu 40
<210> 521
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> CSH-tgt3_3'_d10
<400> 521
taauuucuac ucutguagau gguguacagc aguggcuggu 40
<210> 522
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> CSH-tgt3_3'_d11
<400> 522
taauuucuac ucutguagau gguguacagc aguggcuggu 40
<210> 523
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> CSH-tgt3_3'_d12
<400> 523
taauuucuac ucutguagau gguguacagc aguggcuggu 40
<210> 524
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> CSH-tgt3_3'_d14
<400> 524
taauuucuac ucutguagau gguguacagc aguggcuggu 40
<210> 525
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> CSH-tgt3_3'_d15
<400> 525
taauuucuac ucutguagau gguguacagc aguggcuggu 40
<210> 526
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> CSH-tgt3_3'_d17
<400> 526
taauuucuac ucutguagau gguguacagc aguggctggu 40
<210> 527
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> CSH-tgt3_3'_d18
<400> 527
taauuucuac ucutguagau gguguacagc aguggcuggu 40
<210> 528
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> CSH-tgt3_3'_d19
<400> 528
taauuucuac ucutguagau gguguacagc aguggcuggu 40
<210> 529
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
oligonucleotide
<220>
<223> Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide
<220>
<223> CSH-tgt3_3'_d20
<400> 529
taauuucuac ucutguagau gguguacagc aguggcuggt 40
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| КОМПОЗИЦИИ ОЛИГОНУКЛЕОТИДОВ И СПОСОБЫ С НИМИ | 2016 |
|
RU2830607C2 |
| БИСПЕЦИФИЧЕСКИЕ АНТИГЕНСВЯЗЫВАЮЩИЕ КОНСТРУКЦИИ ПРОТИВ HER2 | 2014 |
|
RU2737882C2 |
| КОМПОНЕНТЫ СИСТЕМЫ CRISPR-CAS, СПОСОБЫ И КОМПОЗИЦИИ ДЛЯ МАНИПУЛЯЦИИ С ПОСЛЕДОВАТЕЛЬНОСТЯМИ | 2013 |
|
RU2796549C2 |
| КОНСТРУИРОВАНИЕ СИСТЕМ, СПОСОБЫ И ОПТИМИЗИРОВАННЫЕ НАПРАВЛЯЮЩИЕ КОМПОЗИЦИИ ДЛЯ МАНИПУЛЯЦИИ С ПОСЛЕДОВАТЕЛЬНОСТЯМИ | 2013 |
|
RU2796017C2 |
| Антитела против CXCR2 и их применение | 2019 |
|
RU2807067C2 |
| ДОСТАВКА, КОНСТРУИРОВАНИЕ И ОПТИМИЗАЦИЯ СИСТЕМ, СПОСОБОВ И КОМПОЗИЦИЙ ДЛЯ МАНИПУЛЯЦИИ С ПОСЛЕДОВАТЕЛЬНОСТЯМИ И ПРИМЕНЕНИЯ В ТЕРАПИИ | 2013 |
|
RU2721275C2 |
| АНТИТЕЛА ПРОТИВ PD-L1 И ИХ ВАРИАНТЫ | 2017 |
|
RU2770590C2 |
| НАПРАВЛЯЕМАЯ РНК РЕГУЛЯЦИЯ ТРАНСКРИПЦИИ | 2014 |
|
RU2756865C2 |
| НОВЫЕ ФЕРМЕНТЫ И СИСТЕМЫ CRISPR | 2016 |
|
RU2777988C2 |
| СИСТЕМЫ CRISPR-CAS И СПОСОБЫ ИЗМЕНЕНИЯ ЭКСПРЕССИИ ПРОДУКТОВ ГЕНОВ | 2013 |
|
RU2796273C2 |
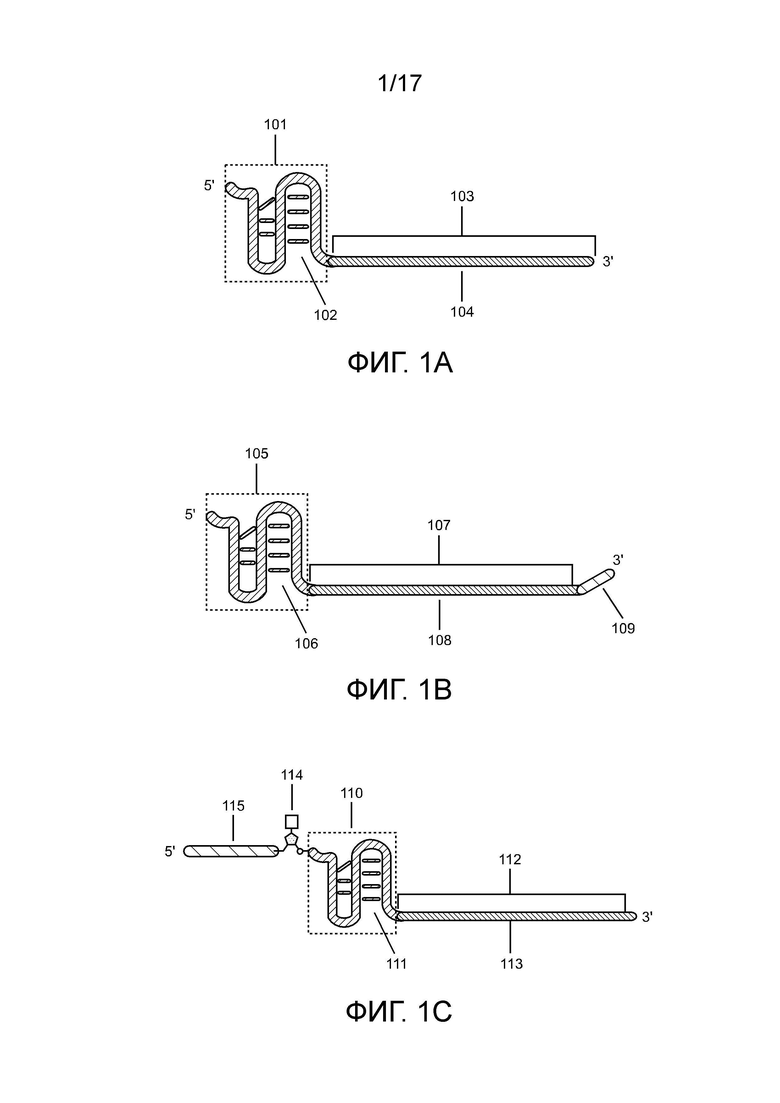
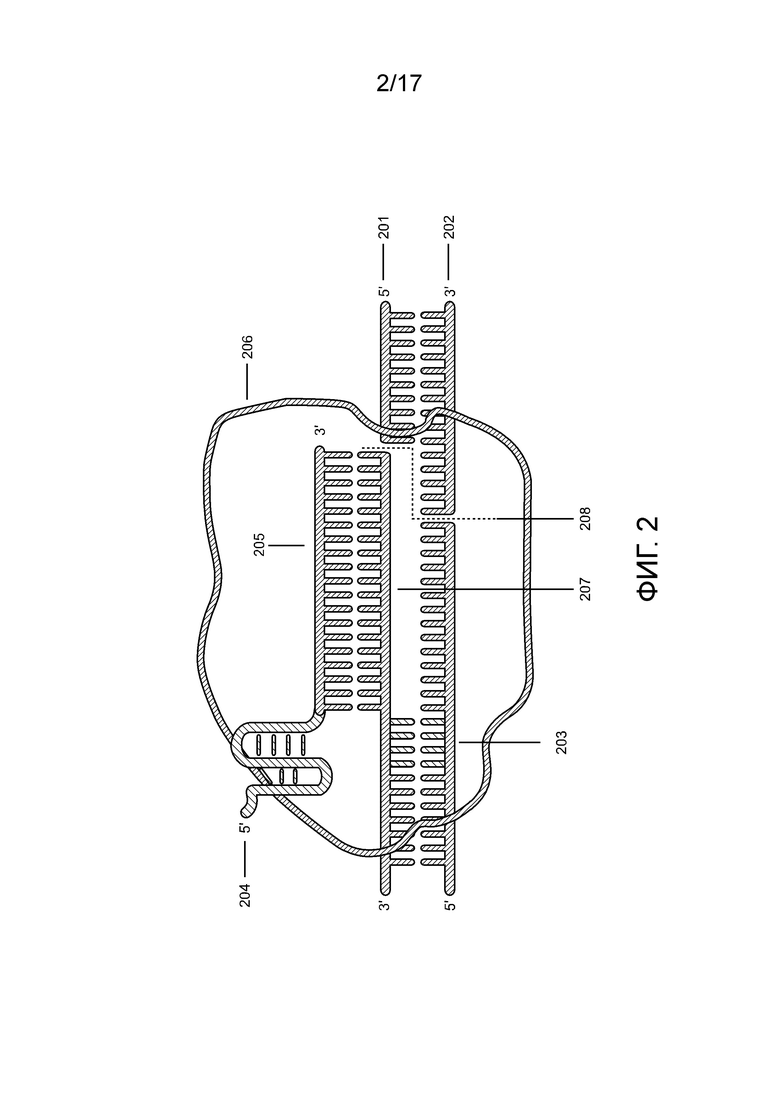
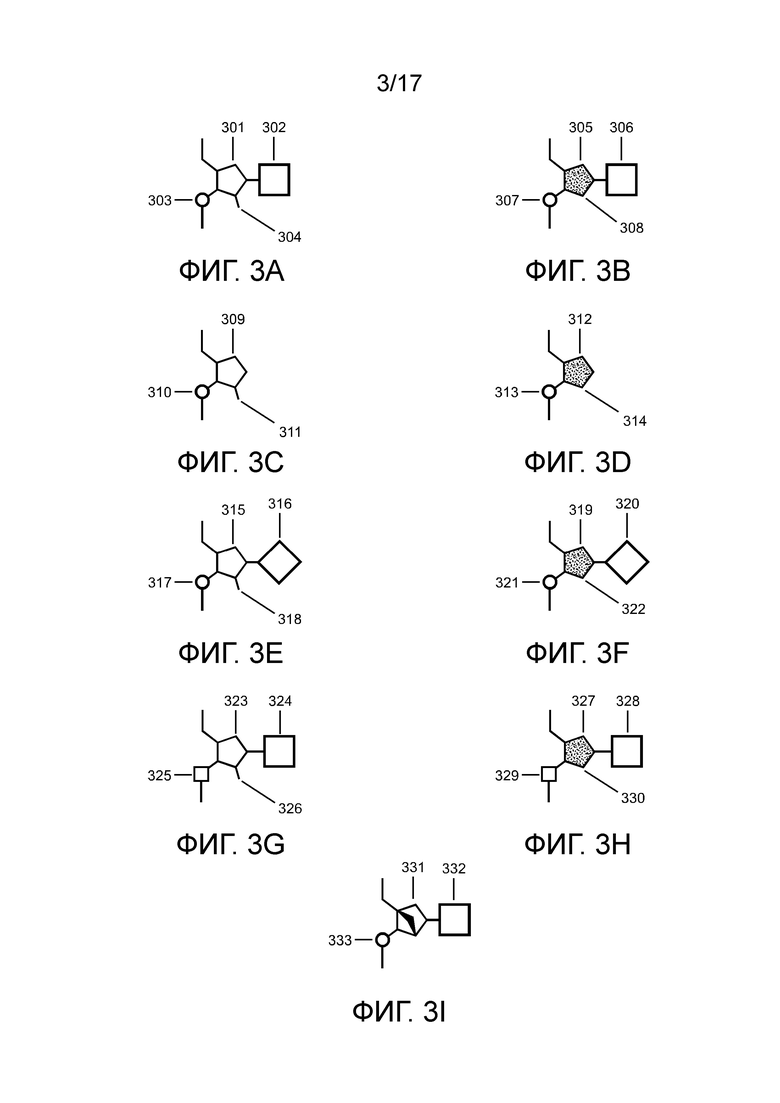
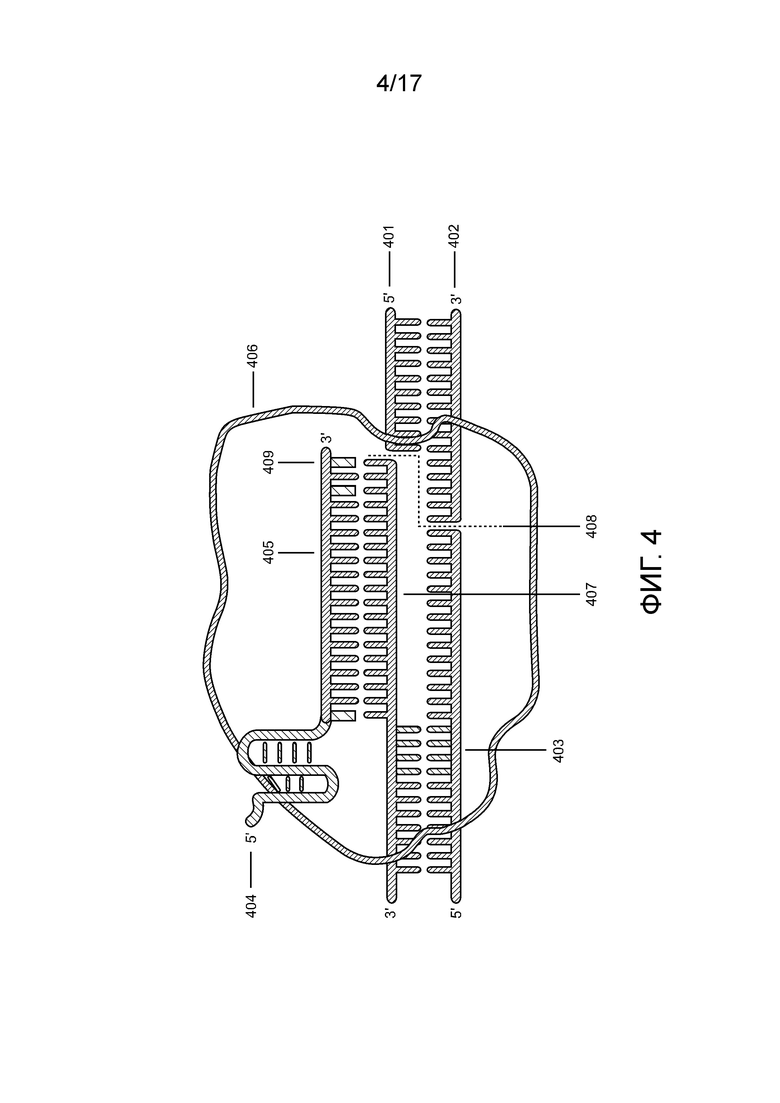
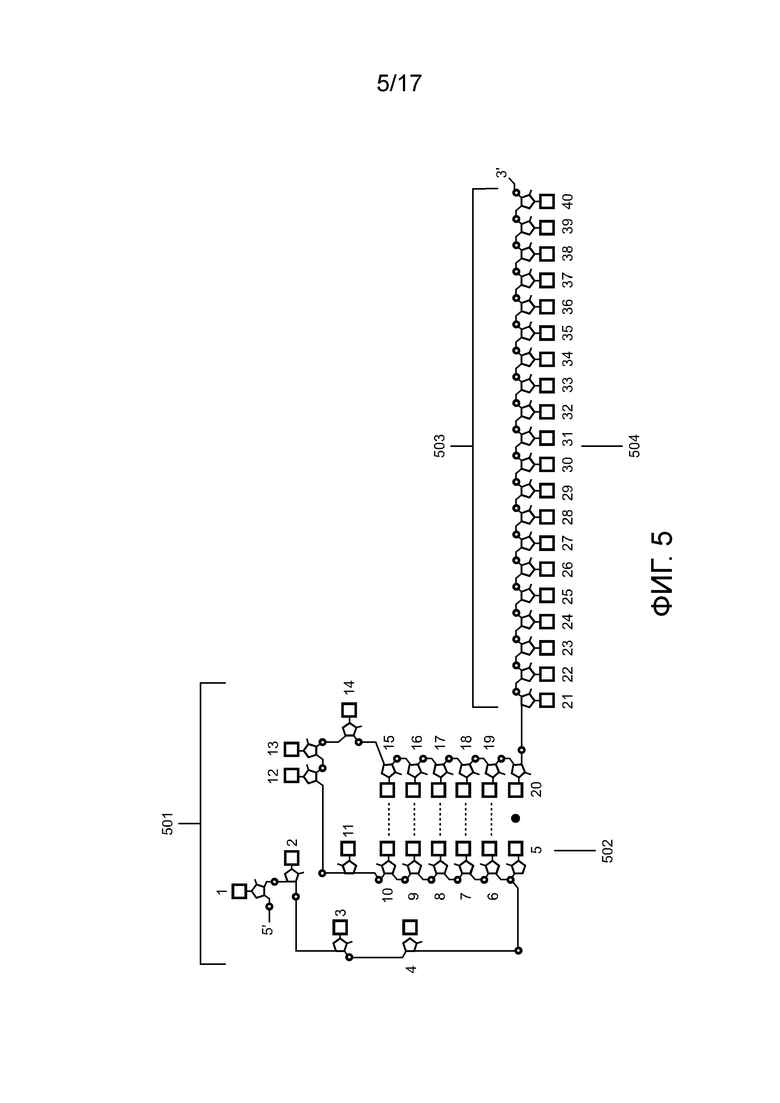
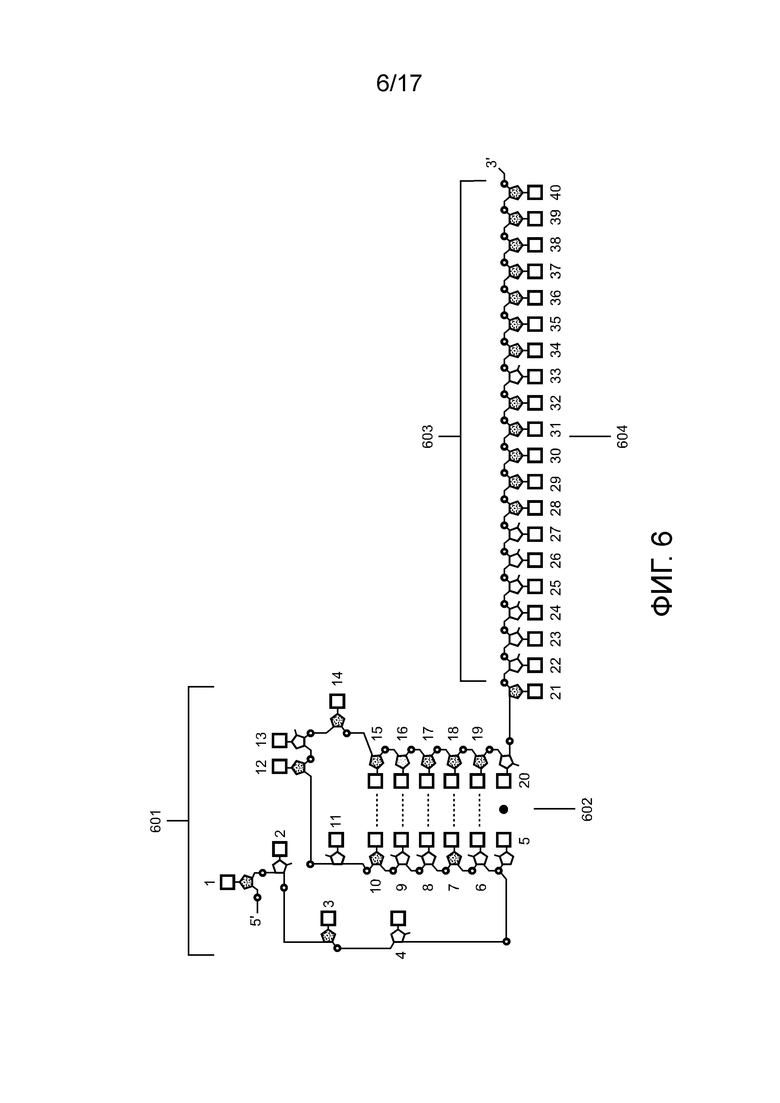
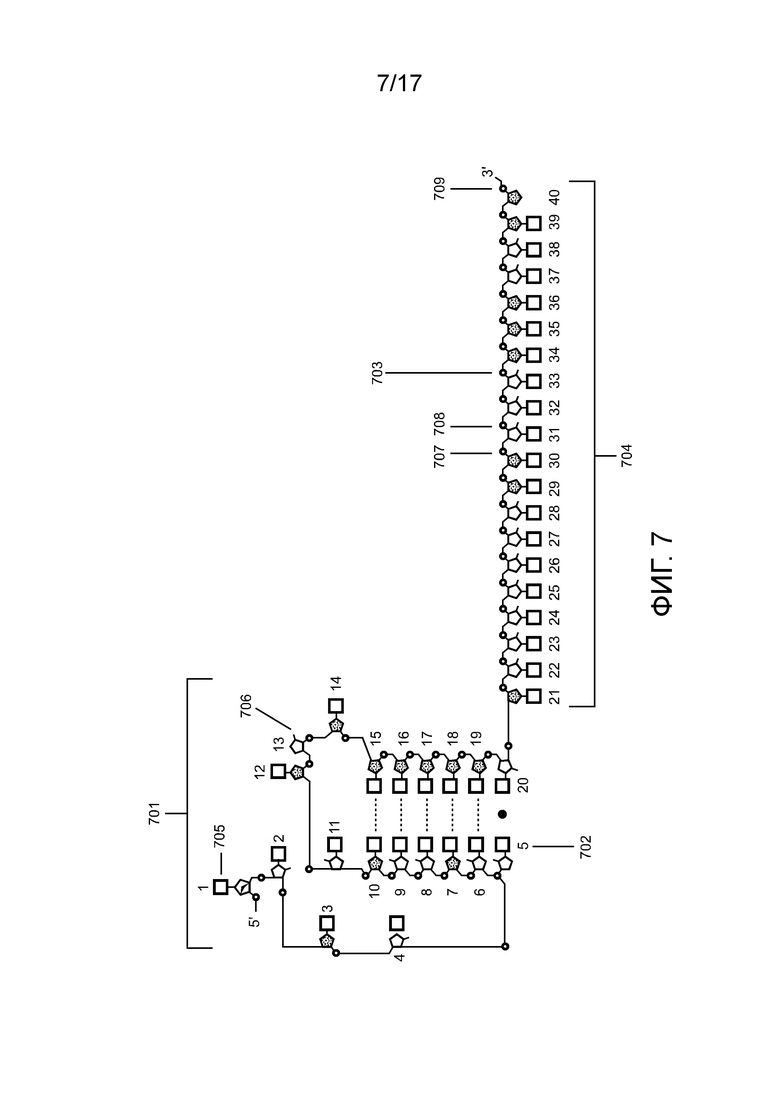
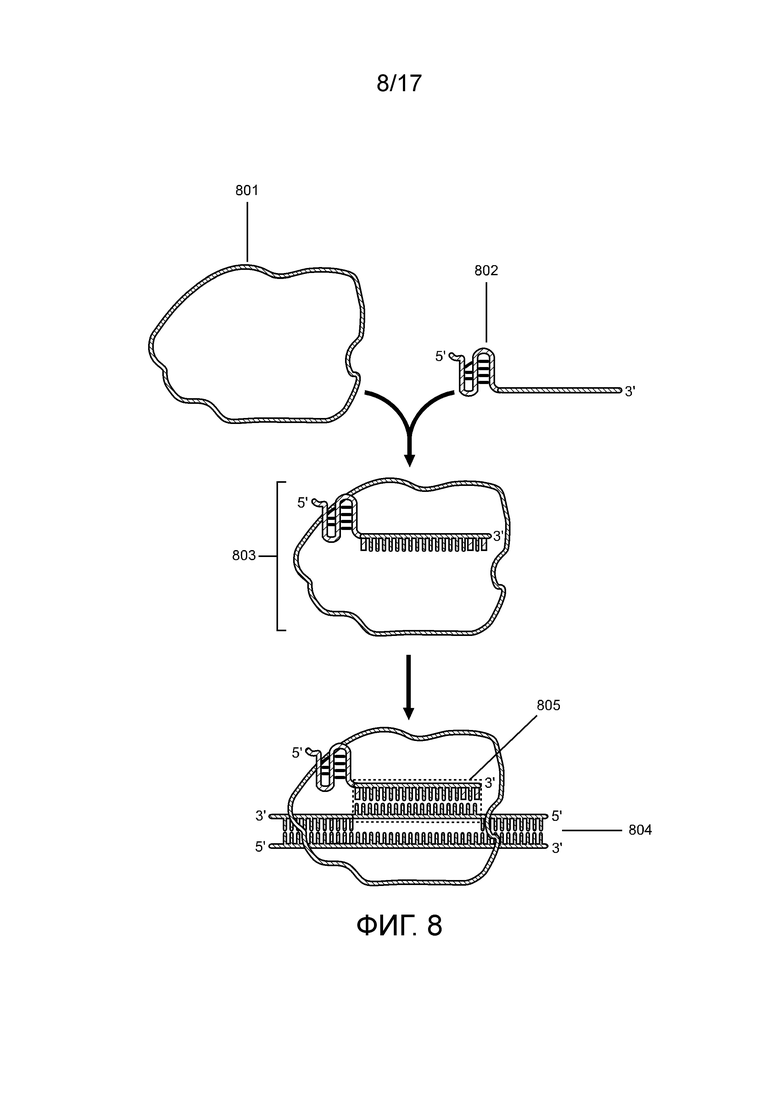
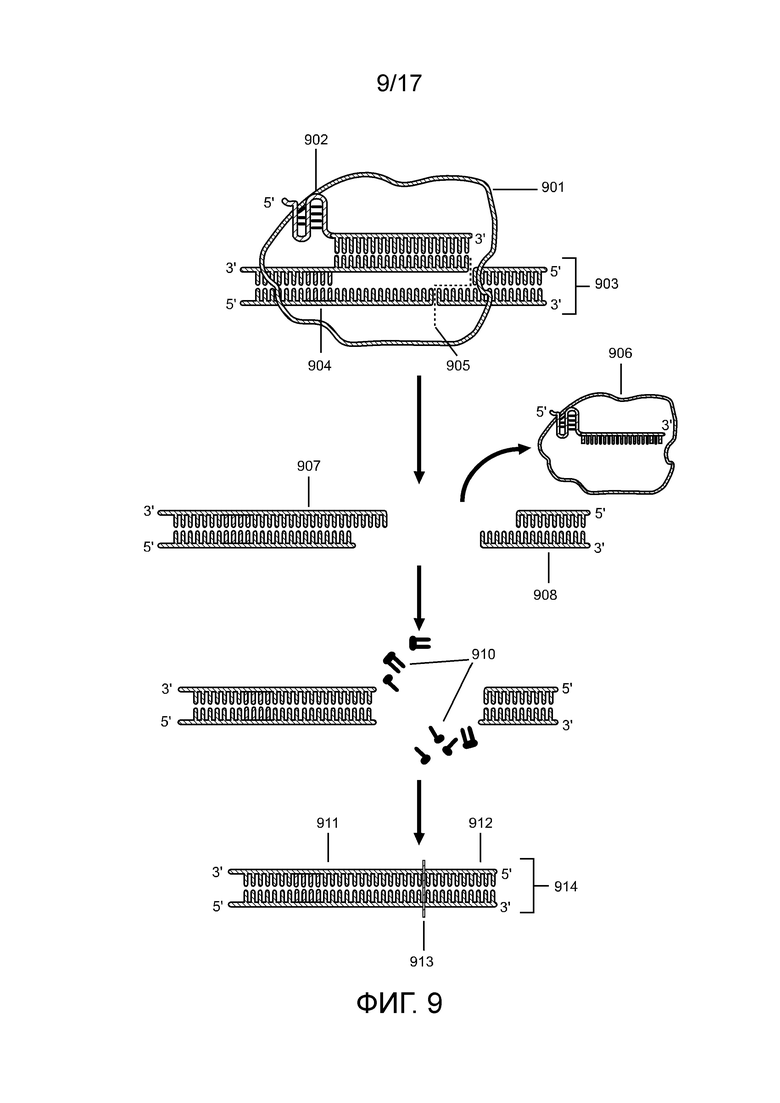
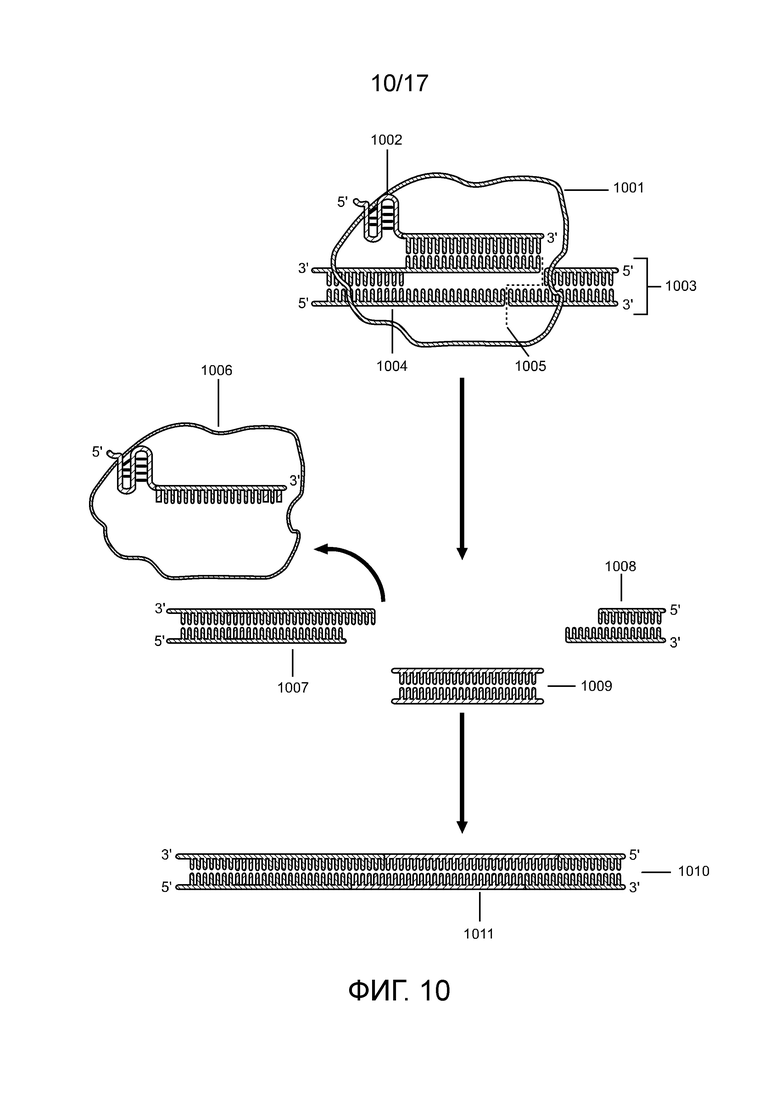
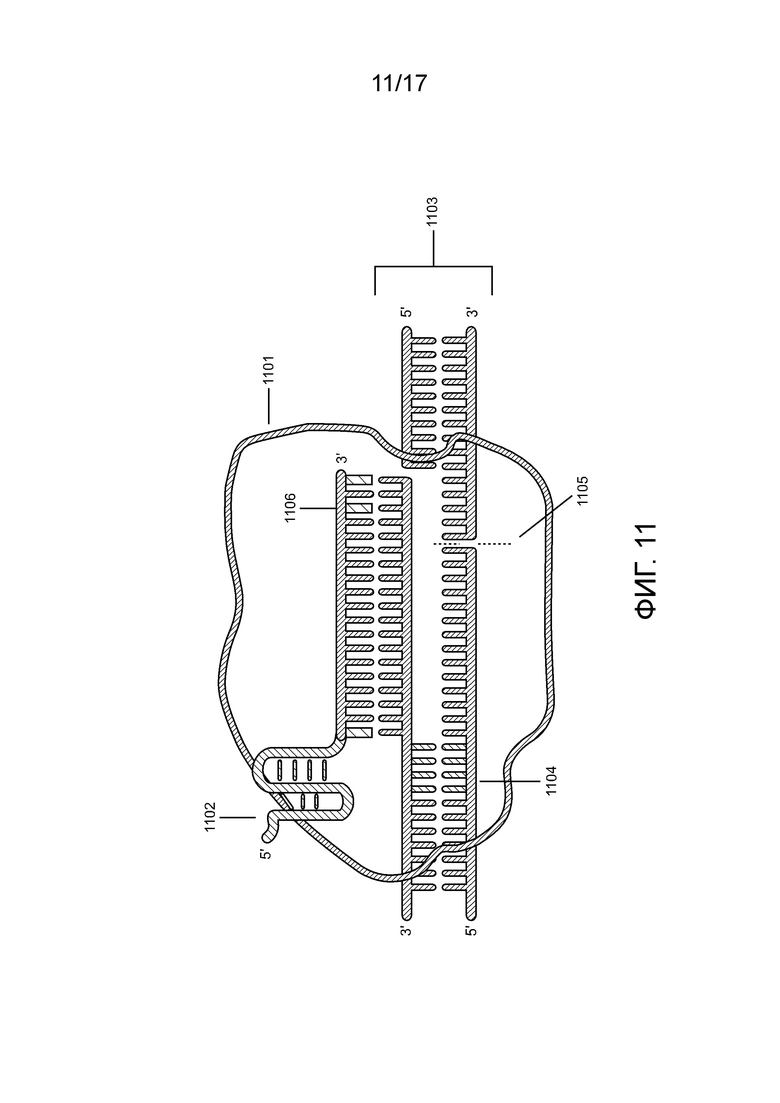
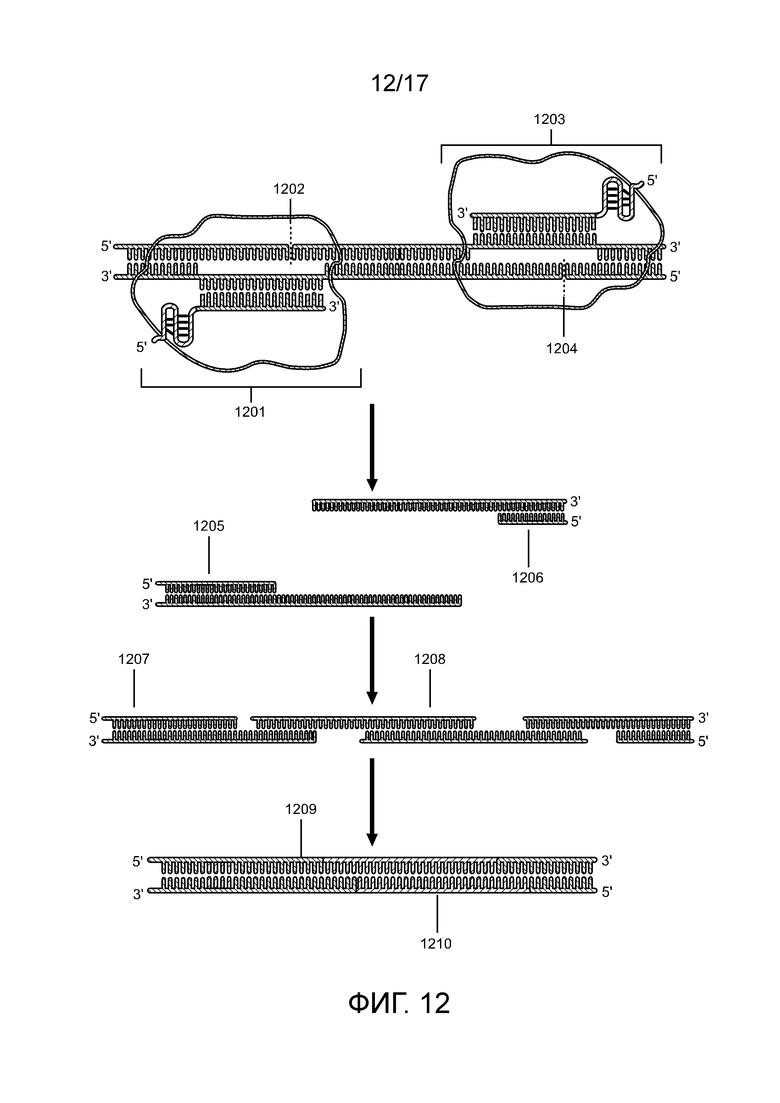
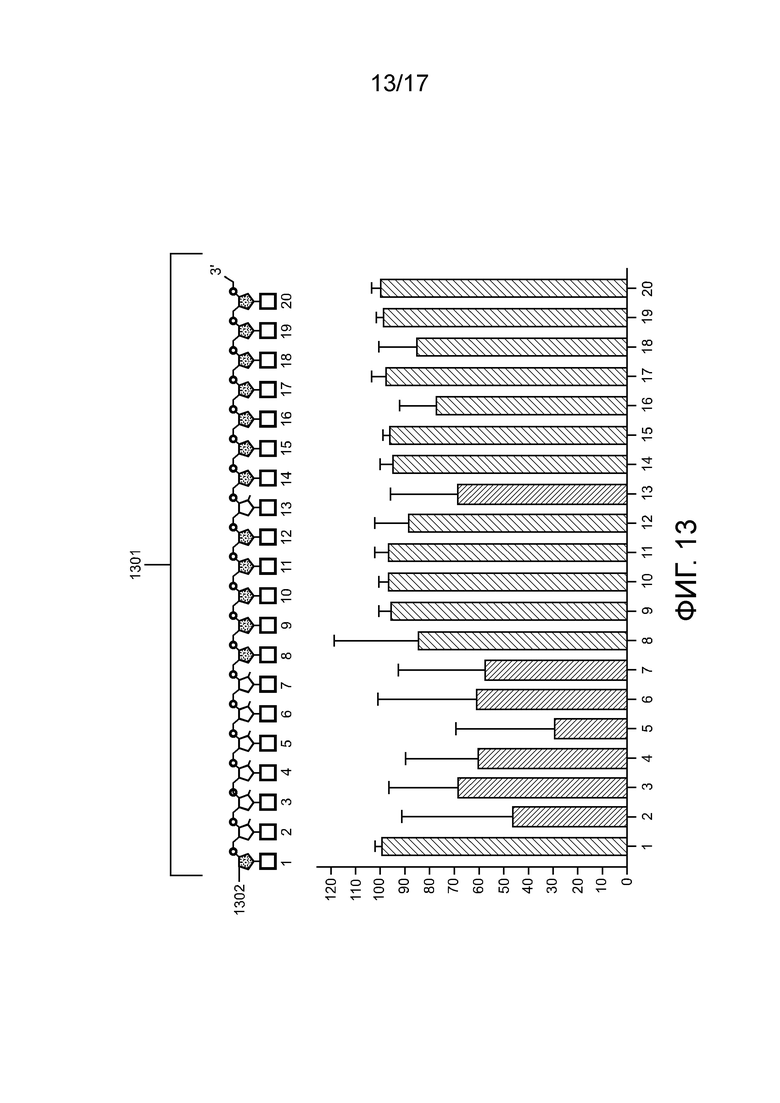
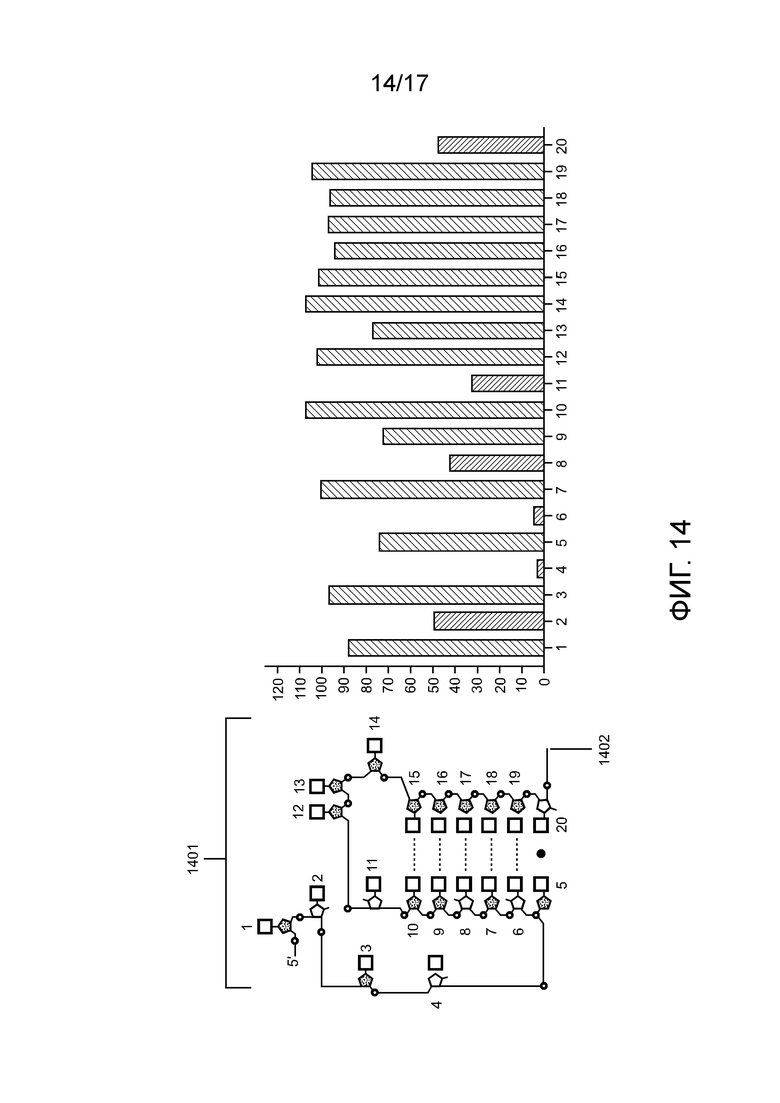
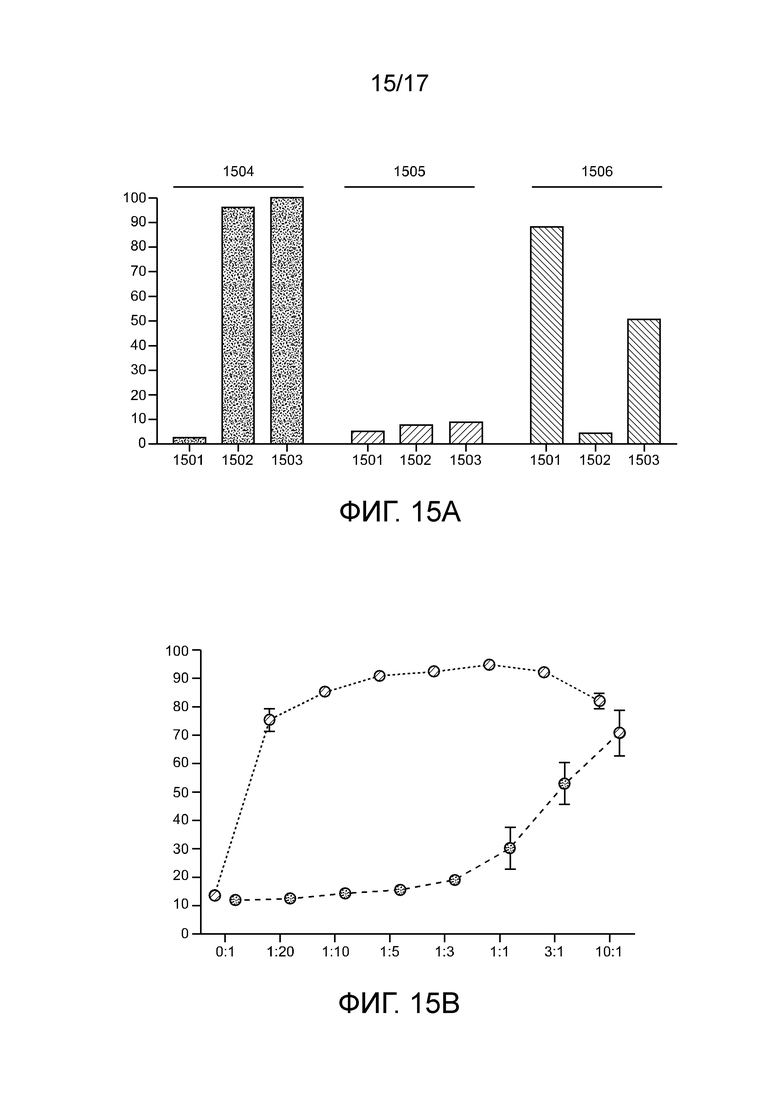
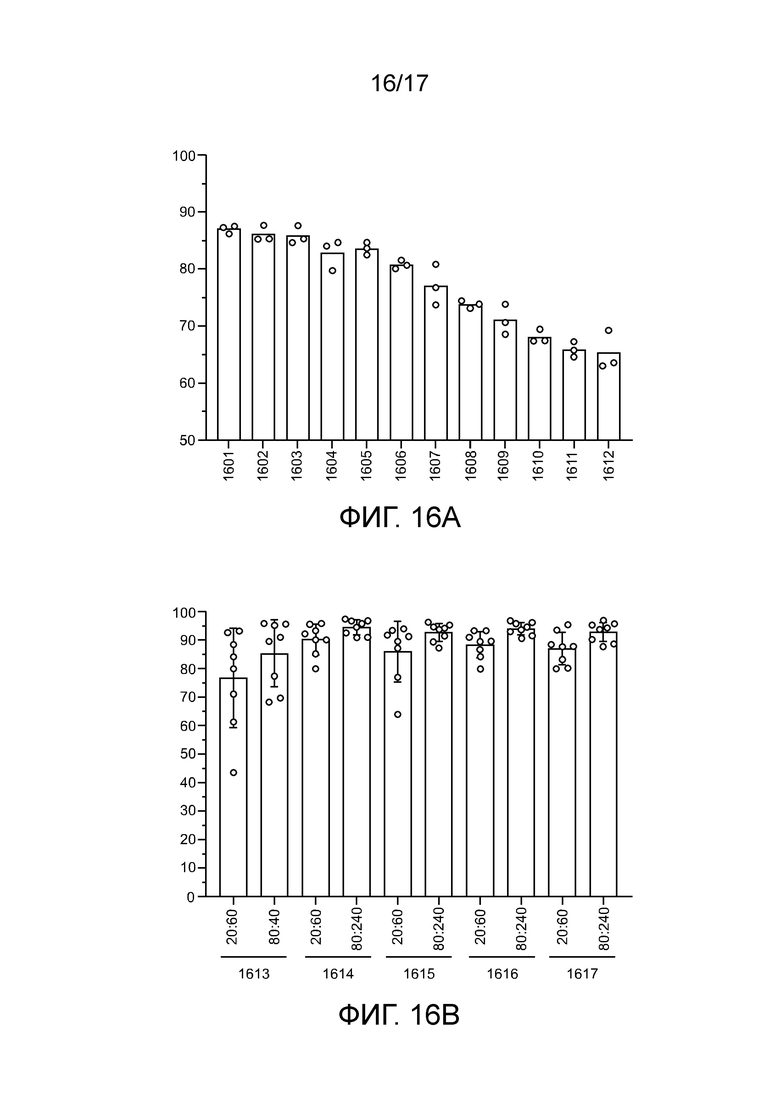
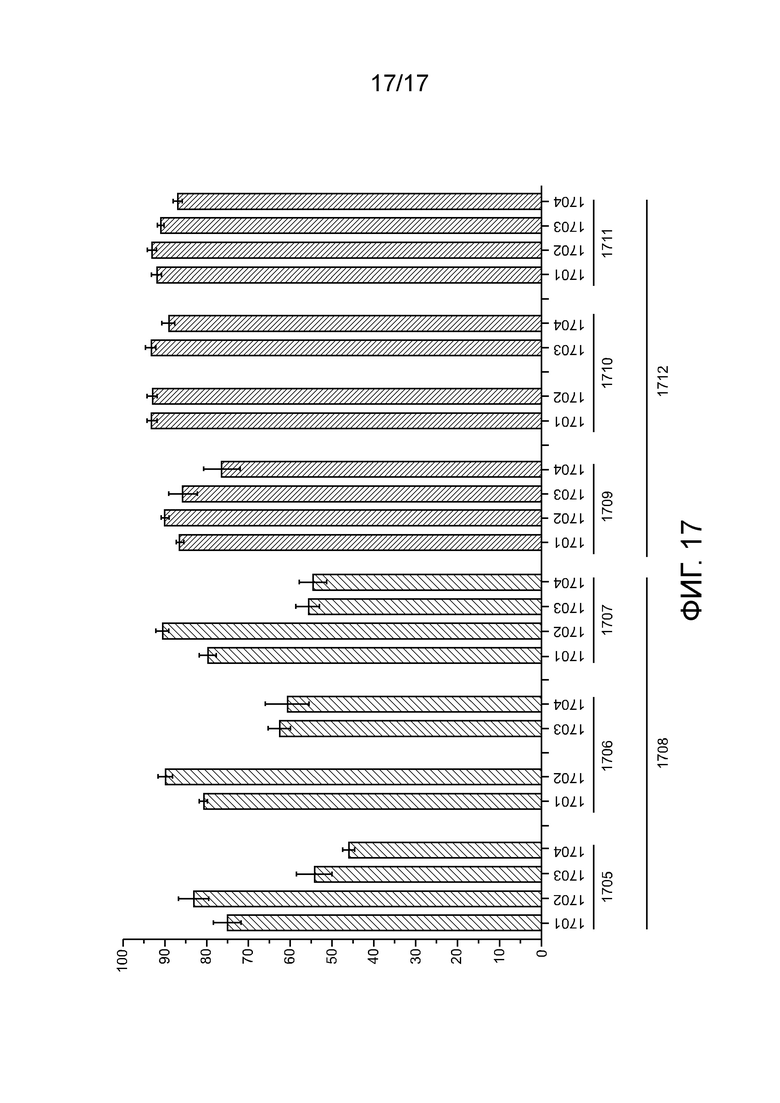
Изобретение относится к области биотехнологии, а именно к способу получения клетки, экспрессирующей химерный антигенный рецептор (CAR), включающему приведение первой нуклеиновой кислоты-мишени, содержащей последовательность TRAC, в клетке в контакт с первой композицией нуклеиновой кислоты/белка CRISPR, содержащей направляющую молекулу CRISPR и белок Cas12, и приведение второй последовательности нуклеиновой кислоты-мишени, содержащей последовательность В2М, в той же клетке в контакт со второй композицией нуклеиновой кислоты/белка CRISPR, содержащей направляющую молекулу CRISPR и белок Cas12, а также к CAR-экспрессирующей клетке, полученной указанным способом. Изобретение эффективно для получения CAR-экспрессирующей клетки для адоптивной клеточной терапии. 3 н. и 17 з.п. ф-лы, 17 ил., 29 табл., 14 пр.
1. Способ получения клетки, экспрессирующей химерный антигенный рецептор (CAR), где указанный способ включает:
a) приведение первой нуклеиновой кислоты-мишени, содержащей последовательность TRAC, в клетке в контакт с первой композицией нуклеиновой кислоты/белка CRISPR, содержащей направляющую молекулу CRISPR и белок Cas12, где направляющая молекула CRISPR содержит нацеливающую область, способную связываться с последовательностью нуклеиновой кислоты-мишени, и активирующую область, содержащую последовательность РНК UAAUUUCUACUCUUGUAGAU (SEQ ID NO: 6), включающую по меньшей мере один дезоксирибонуклеотид вместо рибонуклеотида, где активирующая область способна образовывать нуклеопротеиновый комплекс с белком Cas12, где указанная первая композиция нуклеиновой кислоты/белка CRISPR способна расщеплять первую последовательность нуклеиновой кислоты-мишени;
b) приведение второй последовательности нуклеиновой кислоты-мишени, содержащей последовательность В2М, в той же клетке в контакт со второй композицией нуклеиновой кислоты/белка CRISPR, содержащей направляющую молекулу CRISPR и белок Cas12, где направляющая молекула CRISPR содержит нацеливающую область, способную связываться с последовательностью нуклеиновой кислоты-мишени, и активирующую область, содержащую последовательность РНК UAAUUUCUACUCUUGUAGAU (SEQ ID NO: 6), включающую по меньшей мере один дезоксирибонуклеотид вместо рибонуклеотида, где активирующая область способна образовывать нуклеопротеиновый комплекс с белком Cas12, где указанная вторая композиция нуклеиновой кислоты/белка CRISPR способна расщеплять вторую последовательность нуклеиновой кислоты-мишени;
c) получение первого донорного полинуклеотида, кодирующего CAR, содержащий scFv, трансмембранный домен, костимулирующий домен и активирующий домен, где CAR способен встраиваться в сайт расщепления в первой последовательности нуклеиновой кислоты-мишени;
d) получение второго донорного полинуклеотида, кодирующего конструкцию слияния B2M-HLA-E, содержащую сигнал секреции В2М, сигнальную последовательность пептида HLA-G, первую линкерную последовательность, последовательность В2М, вторую линкерную последовательность и последовательность HLA-E, где конструкция слияния B2M-HLA-E способна встраиваться в сайт расщепления во второй последовательности нуклеиновой кислоты-мишени;
e) расщепление первой последовательности нуклеиновой кислоты-мишени и встраивание по меньшей мере части первого донорного полинуклеотида в сайт расщепления; и
f) расщепление второй последовательности нуклеиновой кислоты-мишени и встраивание по меньшей мере части второго донорного полинуклеотида в сайт расщепления.
2. Способ по п. 1, где одно или более положений 1, 3, 7, 10, 12, 14, 15, 16, 17, 18 и 19 в активирующей области содержат дезоксирибонуклеотидное основание.
3. Способ по п. 1, где активирующая область содержит последовательность, выбранную из SEQ ID NO: 459-465.
4. Способ по п. 1, где направляющая молекула CRISPR содержит одну или более фосфортиоатных химических модификаций между последними 3-5 нуклеотидами на 5'-конце или 3'-конце полинуклеотидной последовательности.
5. Способ по п. 4, где направляющая молекула CRISPR содержит последовательность, выбранную из SEQ ID NO: 512-517.
6. Способ по п. 1, где нацеливающая область нацелена на ген В2М и содержит последовательность РНК AGUGGGGGUGAAUUCAGUGU (SEQ ID NO: 211), где одно или более положений 1, 8, 9, 10, 11, 12, 14, 15, 16, 17, 18, 19 и 20 в нацеливающей области содержат дезоксирибонуклеотидное основание.
7. Способ по п. 6, где направляющая молекула CRISPR содержит последовательность, выбранную из SEQ ID NO: 212-231, 275-294, 296-315, 331-334 и 346-350.
8. Способ по п. 6, где направляющая молекула CRISPR содержит последовательность SEQ ID NO: 416.
9. Способ по п. 1, где нацеливающая область нацелена на ген TRAC и содержит последовательность РНК GAGUCUCUCAGCUGGUACAC (SEQ ID NO: 232), где одно или более положений 1, 8, 9, 10, 11, 12, 14, 15, 16, 17, 18, 19 и 20 в нацеливающей области содержат дезоксирибонуклеотидное основание.
10. Способ по п. 9, где направляющая молекула CRISPR содержит последовательность, выбранную из SEQ ID NO: 233-252, 317-329, 331-334, 491-492 и 508.
11. Способ по п. 9, где направляющая молекула CRISPR содержит последовательность SEQ ID NO: 415.
12. Способ по п. 1, где нацеливающая область нацелена на ген CISH и содержит последовательность РНК, способную к гибридизации с человеческой геномной последовательностью, выбранной из SEQ ID NO: 156-165, где одно или более положений 1, 8, 9, 10, 11, 12, 14, 15, 16, 17, 18, 19 и 20 в нацеливающей области содержат дезоксирибонуклеотидное основание.
13. Способ по п. 1, где нацеливающая область нацелена на ген PDCD1 и содержит последовательность РНК, способную к гибридизации с человеческой геномной последовательностью, выбранной из SEQ ID NO: 134-155, где одно или более положений 1, 8, 9, 10, 11, 12, 14, 15, 16, 17, 18, 19 и 20 в нацеливающей области содержат дезоксирибонуклеотидное основание.
14. Способ по п. 1, где нацеливающая область нацелена на ген CBLB и содержит последовательность РНК, способную к гибридизации с человеческой геномной последовательностью, выбранной из SEQ ID NO: 166-189, где одно или более положений 1, 8, 9, 10, 11, 12, 14, 15, 16, 17, 18, 19 и 20 в нацеливающей области содержат дезоксирибонуклеотидное основание.
15. Способ по п. 1, где белок Casl2 представляет собой белок Cas12a, содержащий у С-конца последовательность, которая содержит линкер и сигнал ядерной локализации (NLS) и которая по меньшей мере на 80% идентична аминокислотной последовательности, выбранной из группы, состоящей из SEQ ID NO: 479-490.
16. Способ по п. 15, где белок Cas12a содержит аминокислотную последовательность SEQ ID NO: 1.
17. Способ по п. 1, где второй донорный полинуклеотид содержит SEQ ID NO: 414.
18. Способ по п. 1, где CAR содержит трансмембранный домен, происходящий от CD8, костимулирующий домен 4-1 ВВ или костимулирующий домен CD28 и активирующий домен CD3ζ.
19. CAR-экспрессирующая клетка, полученная способом по п. 1, содержащая последовательность SEQ ID NO: 414, где клетку выбирают из лимфоцита, CAR-T-клетки, TCR-клетки, CAR-T-клетки, сконструированной на основе TCR, TIL, CAR-TIL, дендритной клетки, CAR-DC, макрофага, CAR-M, клетки iPSC, клетки, дифференцированной из iPSC, NK-клетки или CAR-NK-клетки.
20. Способ адоптивной клеточной терапии, включающий введение индивидууму, нуждающемуся в этом, CAR-экспрессирующей клетки по п. 19.
| WO 2017218185 A1, 21.12.2017 | |||
| WO 2017190664 A1, 09.11.2017 | |||
| WO 2018009822 A1, 11.01.2018 | |||
| MARIA PALLARES MASMITJA et al., CRISPR-gRNA Design, Yonglun Luo (ed.), CRISPR Gene Editing: Methods and Protocols, Methods in Molecular Biology, 2019, vol | |||
| Судно | 1925 |
|
SU1961A1 |
| RU 2017127051 A, 01.02.2019. | |||
Авторы
Даты
2024-12-23—Публикация
2021-10-18—Подача