Родственные заявки и включение при помощи ссылки
Данная заявка заявляет приоритет предварительной заявки на патент США 61/842322 и заявки на патент США 14/054414, каждая с общей ссылкой BI-2011/008A, озаглавленные "СИСТЕМЫ CRISPR-Cas И СПОСОБЫ ИЗМЕНЕНИЯ ЭКСПРЕССИИ ПРОДУКТОВ ГЕНОВ" (CRISPR-CAS SYSTEMS AND METHODS FOR ALTERING EXPRESSION OF GENE PRODUCTS), поданных 2 июля 2013 г. и 15 октября 2013 г., соответственно. Также заявляется приоритет предварительных заявок на патент США 61/736527, 61/748427, 61/791409 и 61/835931 с общей ссылкой BI-2011/008/WSGR, номер в реестре 44063-701.101, BI-2011/008/WSGR, номер в реестре 44063-701.102, общей ссылкой BI-2011/008/VP, номер в реестре 44790.02.2003 и BI-2011/008/VP, номер в реестре 44790.03.2003, соответственно, все из которых озаглавлены "СИСТЕМЫ, СПОСОБЫ И КОМПОЗИЦИИ ДЛЯ МАНИПУЛЯЦИИ С ПОСЛЕДОВАТЕЛЬНОСТЯМИ" (SYSTEMS METHODS AND COMPOSITIONS FOR SEQUENCE MANIPULATION), поданных 12 декабря 2012 г., 2 января 2013 г., 15 марта 2013 г. и 17 июня 2013 г., соответственно.
Делается ссылка на предварительные заявки на патент США 61/758468; 61/769046; 61/802174; 61/806375; 61/814263; 61/819803 и 61/828130, каждая из которых озаглавлена "КОНСТРУИРОВАНИЕ И ОПТИМИЗАЦИЯ СИСТЕМ, СПОСОБОВ И КОМПОЗИЦИЙ ДЛЯ МАНИПУЛЯЦИИ С ПОСЛЕДОВАТЕЛЬНОСТЯМИ" (ENGINEERING AND OPTIMIZATION OF SYSTEMS, METHODS AND COMPOSITIONS FOR SEQUENCE MANIPULATION), поданные 30 января 2013 г.; 25 февраля 2013 г.; 15 марта 2013 г.; 28 марта 2013 г.; 20 апреля 2013 г.; 6 мая 2013 г. и 28 мая 2013 г., соответственно. Также делается ссылка на предварительные заявки на патент США 61/835936, 61/836127, 61/836101, 61/836080 и 61/835973, каждая из которых подана 17 июня 2013 г.
Вышеупомянутые заявки и все документы, упомянутые в них или во время их делопроизводства (“упомянутые в заявке документы”), и все документы, упомянутые или на которые ссылаются в упомянутых в заявке документах, и все документы, упомянутые или на которые ссылаются в данном документе (“документы, упомянутые в данном документе”), и все документы, упомянутые или на которые ссылаются в документах, упомянутых в данном документе, наравне с любыми инструкциями производителя, описаниями, характеристиками продуктов и описаниями продуктов для любых продуктов, упомянутых в данном документе или в любом документе, включенном при помощи ссылки в данный документ, таким образом, включены в данный документ при помощи ссылки и могут быть использованы при осуществлении на практике настоящего изобретения. Более конкретно, все документы, на которые ссылаются, включены при помощи ссылки в такой же мере, как если бы конкретно и отдельно было указано, что каждый отдельный документ включен при помощи ссылки.
Область техники
Настоящее изобретение в целом относится к системам, способам и композициям, применяемым для контроля экспрессии генов, включающего целенаправленное воздействие на последовательность, такое как внесение изменений в геном или редактирование гена, при котором можно использовать векторные системы, близкие к коротким палиндромным повторам, регулярно расположенным группами, (CRISPR) и их компонентам.
Утверждение касательно финансируемого из федерального бюджета исследования
Настоящее изобретение было разработано при правительственной поддержке согласно NIH Pioneer Award DP1MH100706, выданному национальными институтами здравоохранения. Правительство обладает определенными правами на настоящее изобретение.
Предпосылки изобретения
Недавние достижения в технологиях секвенирования генома и способах анализа значительно ускорили возможность каталогизации и картирования генетических факторов, ассоциированных с широким разнообразием биологических функций и заболеваний. Технологии точного целенаправленного воздействия на геном необходимы для обеспечения систематичного обратного конструирования казуальных генетических изменений путем обеспечения возможности селективного внесения изменений в отдельные генетические элементы, а также для продвижения применений в области синтетической биологии, биотехнологии и медицины. Несмотря на то, что технологии редактирования генома, такие как оригинальные ферменты с "цинковыми пальцами", подобные транскрипционным активаторам эффекторы (TALE) или хоминг-мегануклеазы, доступны для осуществления целенаправленного внесения изменений в геном, все еще существует необходимость в новых технологиях конструирования генома, которые являются доступными, простыми в осуществлении, масштабируемыми и характеризуются возможностью целенаправленного воздействия на несколько положений в эукариотическом геноме.
Краткое описание изобретения
Существует актуальная необходимость в альтернативных и функциональных системах и технологиях для целенаправленного воздействия на последовательность с широким спектром применений. Настоящее изобретение удовлетворяет этой необходимости и предусматривает связанные с этим преимущества. CRISPR/Cas или система CRISPR-Cas (оба выражения используют взаимозаменяемо по всей данной заявке) не предусматривает получение индивидуализированных белков для целенаправленного воздействия на конкретные последовательности, но скорее один фермент Cas может быть запрограммирован короткой молекулой РНК для узнавания специфичной ДНК-мишени, другими словами, фермент Cas может связываться со специфичной ДНК-мишенью при помощи указанной короткой молекулы РНК. Добавление системы CRISPR-Cas к спектру технологий секвенирования генома и способов анализа может значительно упростить методику и ускорить возможность каталогизации и картирования генетических факторов, ассоциированных с широким разнообразием биологических функций и заболеваний. Для того, чтобы эффективно использовать систему CRISPR-Cas для редактирования генома без вредного действия, важно понимать аспекты конструирования и оптимизации этих средств для конструирования генома, которые являются аспектами заявленного изобретения.
В одном аспекте настоящее изобретение предусматривает способ изменения или модификации экспрессии одного или нескольких продуктов генов, который может включать введение в клетку, содержащую и экспрессирующую молекулы ДНК, кодирующие один или несколько продуктов генов, сконструированной не встречающейся в природе системы CRISPR-Cas, которая может содержать белок Cas и одну или несколько направляющих РНК, которые осуществляют нацеливание на молекулы ДНК, вследствие чего одна или несколько направляющих РНК осуществляют нацеливание на геномные локусы молекул ДНК, кодирующих один или несколько продуктов генов, а белок Cas расщепляет геномные локусы молекул ДНК, кодирующих один или несколько продуктов генов, вследствие чего экспрессия одного или нескольких продуктов генов изменяется или модифицируется; и при этом белок Cas и направляющая РНК не встречаются вместе в естественных условиях. Настоящее изобретение предполагает, что экспрессия двух или более продуктов генов может быть изменена или модифицирована. Настоящее изобретение также предполагает, что направляющая РНК содержит направляющую последовательность, слитую с tracr-последовательностью. В предпочтительном варианте осуществления клетка является эукариотической клеткой, в более предпочтительном варианте осуществления клетка является клеткой млекопитающего и в еще более предпочтительном варианте осуществления клетка млекопитающего является клеткой человека. Настоящее изобретение также предполагает, что белок Cas может содержать один или несколько сигналов ядерной локализации (NLS). В некоторых вариантах осуществления белок Cas является ферментом системы CRISPR II типа. В некоторых вариантах осуществления белок Cas является белком Cas9. В некоторых вариантах осуществления белок Cas9 является Cas9 S. pneumoniae, S. pyogenes или S. thermophilus и может включать мутированный Cas9, полученный из этих организмов. Белок может быть гомологом или ортологом Cas9. В некоторых вариантах осуществления белок Cas кодон-оптимизирован для экспрессии в эукариотической клетке. В некоторых вариантах осуществления белок Cas управляет расщеплением одной или двух нитей в определенной точке целевой последовательности. В дополнительном аспекте настоящего изобретения экспрессия продукта гена снижается, и продуктом гена является белок. Настоящее изобретение предполагает, что введение в клетку осуществляют с помощью системы доставки, которая может включать вирусные частицы, липосомы, электропорацию, микроинъекцию или конъюгацию.
В другом аспекте настоящее изобретение предусматривает способ изменения или модификации экспрессии одного или нескольких продуктов генов, включающий введение в клетку, содержащую и экспрессирующую молекулы ДНК, кодирующие один или несколько продуктов генов, сконструированной не встречающейся в природе векторной системы, которая может содержать один или несколько векторов, содержащих a) первый регуляторный элемент, функционально связанный с одной или несколькими направляющими РНК системы CRISPR-Cas, которые гибридизируются с целевыми последовательностями в геномных локусах молекул ДНК, кодирующих один или несколько продуктов генов, b)второй регуляторный элемент, функционально связанный с белком Cas, где компоненты (a) и (b) находятся в одном и том же или разных векторах системы, вследствие чего направляющие РНК осуществляют нацеливание на геномные локусы молекул ДНК, кодирующих один или несколько продуктов генов, а белок Cas расщепляет геномные локусы молекул ДНК, кодирующих один или несколько продуктов генов, вследствие чего экспрессия одного или нескольких продуктов генов изменяется или модифицируется; и где белок Cas и направляющие РНК не встречаются вместе в естественных условиях. Настоящее изобретение предполагает, что экспрессия двух или более продуктов генов может быть изменена или модифицирована. Настоящее изобретение также предполагает, что направляющая РНК содержит направляющую последовательность, слитую с tracr-последовательностью. В предпочтительном варианте осуществления клетка является эукариотической клеткой, в более предпочтительном варианте осуществления клетка является клеткой млекопитающего и в еще более предпочтительном варианте осуществления клетка млекопитающего является клеткой человека. Настоящее изобретение также предполагает, что векторы системы могут дополнительно содержать один или несколько NLS. В некоторых вариантах осуществления белок Cas является ферментом системы CRISPR II типа. В некоторых вариантах осуществления белок Cas является белком Cas9. В некоторых вариантах осуществления белок Cas9 является Cas9 S. pneumoniae, S. pyogenes или S. thermophilus и может включать мутированный Cas9, полученный из этих организмов. Белок может быть гомологом или ортологом Cas9. В некоторых вариантах осуществления белок Cas кодон-оптимизирован для экспрессии в эукариотической клетке. В некоторых вариантах осуществления белок Cas управляет расщеплением одной или двух нитей в определенной точке целевой последовательности. В дополнительном аспекте настоящего изобретения экспрессия продукта гена снижается, и продуктом гена является белок. Настоящее изобретение предполагает, что введение в клетку осуществляют с помощью системы доставки, которая может включать вирусные частицы, липосомы, электропорацию, микроинъекцию или конъюгацию.
Настоящее изобретение также предусматривает сконструированную не встречающуюся в природе векторную систему, которая может содержать один или несколько векторов, содержащих a) первый регуляторный элемент, функционально связанный с одной или несколькими направляющими РНК системы CRISPR-Cas, которые гибридизируются с целевыми последовательностями в геномных локусах молекул ДНК, кодирующих один или несколько продуктов генов, b) второй регуляторный элемент, функционально связанный с белком Cas, где компоненты (a) и (b) находятся в одном и том же или разных векторах системы, вследствие чего направляющие РНК осуществляют нацеливание на геномные локусы молекул ДНК, кодирующих один или несколько продуктов генов, в клетке, а белок Cas расщепляет геномные локусы молекул ДНК, кодирующих один или несколько продуктов генов, вследствие чего экспрессия одного или нескольких продуктов генов изменяется или модифицируется; и где белок Cas и направляющие РНК не встречаются вместе в естественных условиях. Настоящее изобретение предполагает, что экспрессия двух или более продуктов генов может быть изменена или модифицирована. Настоящее изобретение также предполагает, что направляющая РНК содержит направляющую последовательность, слитую с tracr-последовательностью. В предпочтительном варианте осуществления клетка является эукариотической клеткой, в более предпочтительном варианте осуществления клетка является клеткой млекопитающего и в еще более предпочтительном варианте осуществления клетка млекопитающего является клеткой человека. Настоящее изобретение также предполагает, что векторы системы могут дополнительно содержать один или несколько NLS. В некоторых вариантах осуществления белок Cas является ферментом системы CRISPR II типа. В некоторых вариантах осуществления белок Cas является белком Cas9. В некоторых вариантах осуществления белок Cas9 является Cas9 S. pneumoniae, S. pyogenes или S. thermophilus и может включать мутированный Cas9, полученный из этих организмов. Белок может быть гомологом или ортологом Cas9. В некоторых вариантах осуществления белок Cas кодон-оптимизирован для экспрессии в эукариотической клетке. В некоторых вариантах осуществления белок Cas управляет расщеплением одной или двух нитей в определенной точке целевой последовательности. В дополнительном аспекте настоящего изобретения экспрессия продукта гена снижается, и продуктом гена является белок. Настоящее изобретение предполагает, что введение в клетку осуществляют с помощью системы доставки, которая может включать вирусные частицы, липосомы, электропорацию, микроинъекцию или конъюгацию.
В еще одном аспекте настоящее изобретение предусматривает сконструированную программируемую не встречающуюся в природе систему CRISPR-Cas, которая может содержать белок Cas и одну или несколько направляющих РНК, которые осуществляют нацеливание на геномные локусы молекул ДНК, кодирующих один или несколько продуктов генов, в клетке, а белок Cas расщепляет геномные локусы молекул ДНК, кодирующих один или несколько продуктов генов, вследствие чего экспрессия одного или нескольких продуктов генов изменяется или модифицируется; и где белок Cas и направляющие РНК не встречаются вместе в естественных условиях. Настоящее изобретение предполагает, что экспрессия двух или более продуктов генов может изменяться или модифицироваться. Настоящее изобретение также предполагает, что направляющая РНК содержит направляющую последовательность, слитую с tracr-последовательностью. В предпочтительном варианте осуществления клетка является эукариотической клеткой, в более предпочтительном варианте осуществления клетка является клеткой млекопитающего и в еще более предпочтительном варианте осуществления клетка млекопитающего является клеткой человека. Настоящее изобретение также предполагает, что система CRISPR-Cas может дополнительно содержать один или несколько NLS. В некоторых вариантах осуществления белок Cas является ферментом системы CRISPR II типа. В некоторых вариантах осуществления белок Cas является белком Cas9. В некоторых вариантах осуществления белок Cas9 является Cas9 S. pneumoniae, S. pyogenes или S. thermophilus и может включать мутированный Cas9, полученный из этих организмов. Белок может быть гомологом или ортологом Cas9. В некоторых вариантах осуществления белок Cas кодон-оптимизирован для экспрессии в эукариотической клетке. В некоторых вариантах осуществления белок Cas управляет расщеплением одной или двух нитей в определенной точке целевой последовательности. В дополнительном аспекте настоящего изобретения экспрессия продукта гена снижается, и продуктом гена является белок. Настоящее изобретение предполагает, что введение в клетку осуществляют с помощью системы доставки, которая может включать вирусные частицы, липосомы, электропорацию, микроинъекцию или конъюгацию.
В одном аспекте настоящее изобретение предусматривает векторную систему, содержащую один или несколько векторов. В некоторых вариантах осуществления система содержит (a) первый регуляторный элемент, функционально связанный с парной tracr-последовательностью и одним или несколькими сайтами встраивания для встраивания одной или нескольких направляющих последовательностей выше парной tracr-последовательности, где при экспрессии направляющая последовательность управляет специфичным к последовательности связыванием комплекса CRISPR с целевой последовательностью в эукариотической клетке, где комплекс CRISPR содержит фермент CRISPR, образующий комплекс с (1) направляющей последовательностью, которая гибридизируется с целевой последовательностью, и (2) парной tracr-последовательностью, которая гибридизируется с tracr-последовательностью; и (b) второй регуляторный элемент, функционально связанный с кодирующей фермент последовательностью, кодирующей указанный фермент CRISPR, содержащий последовательность ядерной локализации; где компоненты (a) и (b) находятся в одном и том же или в разных векторах системы. В некоторых вариантах осуществления компонент (a) дополнительно содержит tracr-последовательность ниже парной tracr-последовательности под контролем первого регуляторного элемента. В некоторых вариантах осуществления компонент (a) дополнительно содержит две или более направляющие последовательности, функционально связанные с первым регуляторным элементом, где при экспрессии каждая из двух или более направляющих последовательностей управляет специфичным к последовательности связыванием комплекса CRISPR со своей целевой последовательностью в эукариотической клетке. В некоторых вариантах осуществления система содержит tracr-последовательность под контролем третьего регуляторного элемента, такого как промотор полимеразы III. В некоторых вариантах осуществления tracr-последовательность характеризуется по меньшей мере 50%, 60%, 70%, 80%, 90%, 95% или 99% комплементарности последовательности по длине парной tracr-последовательности при оптимальном выравнивании. Определение оптимального выравнивания находится в компетенции специалиста в данной области. Например, существуют публично и коммерчески доступные алгоритмы и программы выравнивания, такие как, без ограничения, ClustalW, Smith-Waterman в matlab, Bowtie, Geneious, Biopython и SeqMan. В некоторых вариантах осуществления комплекс CRISPR содержит одну или несколько последовательностей ядерной локализации, достаточно эффективных, чтобы управлять накоплением указанного комплекса CRISPR в обнаруживаемом количестве в ядре эукариотической клетки. Не желая быть связанными теорией, полагают, что последовательность ядерной локализации не является необходимой для активности комплекса CRISPR у эукариот, но что включение таких последовательностей повышает активность системы, особенно в отношении нацеливания на молекулы нуклеиновых кислот в ядре. В некоторых вариантах осуществления фермент CRISPR является ферментом системы CRISPR II типа. В некоторых вариантах осуществления фермент CRISPR является ферментом Cas9. В некоторых вариантах осуществления фермент Cas9 является Cas9 S. pneumoniae, S. pyogenes или S. thermophilus и может включать мутированный Cas9, полученный из этих организмов. Фермент может быть гомологом или ортологом Cas9. В некоторых вариантах осуществления фермент CRISPR кодон-оптимизирован для экспрессии в эукариотической клетке. В некоторых вариантах осуществления фермент CRISPR управляет расщеплением одной или двух нитей в определенной точке целевой последовательности. В некоторых вариантах осуществления первый регуляторный элемент является промотором полимеразы III. В некоторых вариантах осуществления второй регуляторный элемент является промотором полимеразы II. В некоторых вариантах осуществления направляющая последовательность составляет по меньшей мере 15, 16, 17, 18, 19, 20, 25 нуклеотидов, или от 10 до 30, или от 15 до 25, или от 15 до 20 нуклеотидов в длину. В целом и по всему данному описанию выражение “вектор” относится к молекуле нуклеиновой кислоты, способной переносить другую нуклеиновую кислоту, с которой она была связана. Векторы включают, без ограничения, молекулы нуклеиновых кислот, которые являются одноцепочечными, двухцепочечными или частично двухцепочечными; молекулы нуклеиновых кислот, которые содержат один или несколько свободных концов, не содержат свободных концов (к примеру, кольцевые); молекулы нуклеиновых кислот, которые содержат ДНК, РНК или и ту, и другую; и другие разновидности полинуклеотидов, известных в уровне техники. Одним типом вектора является “плазмида”, которая означает кольцевую петлю двухцепочечной ДНК, в которую можно встраивать дополнительные сегменты ДНК, как, например, при помощи стандартных технологий молекулярного клонирования. Другим типом вектора является вирусный вектор, где полученные из вируса последовательности ДНК или РНК присутствуют в векторе для упаковки в вирус (к примеру, ретровирусы, ретровирусы с дефективной системой репликации, аденовирусы, аденовирусы с дефективной системой репликации и аденоассоциированные вирусы). Вирусные векторы также включают полинуклеотиды, переносимые вирусами для трансфекции клетки-хозяина. Определенные векторы способны к саморегулируемой репликации в клетке-хозяине, в которую они введены (к примеру, бактериальные векторы с бактериальной точкой начала репликации и эписомные векторы млекопитающих). Другие векторы (к примеру, неэписомные векторы млекопитающих) интегрируются в геном клетки-хозяина после введения в клетку-хозяина и, таким образом, реплицируются наряду с геномом хозяина. Более того, определенные векторы способны управлять экспрессией генов, с которыми они функционально связаны. Такие векторы в данном документе называют “векторами экспрессии”. Общепринятые пригодные в технологиях рекомбинантной ДНК векторы экспрессии часто находятся в форме плазмид.
Рекомбинантные векторы экспрессии могут содержать нуклеиновую кислоту согласно настоящему изобретению в форме, подходящей для экспрессии нуклеиновой кислоты в клетке-хозяине, что означает, что рекомбинантные векторы экспрессии включают один или несколько регуляторных элементов, которые могут быть выбраны с учетом клеток-хозяев, которые предполагается использовать для экспрессии, которые функционально связаны с последовательностью нуклеиновой кислоты, экспрессия которой предполагается. В контексте рекомбинантного вектора экспрессии выражение “функционально связанный” предназначено означать, что представляющая интерес нуклеотидная последовательность связана с регуляторным(и) элементом(ами) таким образом, при котором обеспечивается возможность экспрессии нуклеотидной последовательности (к примеру, в in vitro системе транскрипции/трансляции или в клетке-хозяине, если вектор вводят в клетку-хозяина).
Выражение “регуляторный элемент” предназначено включать промоторы, энхансеры, участки внутренней посадки рибосомы (IRES) и другие контролирующие экспрессию элементы (к примеру, сигналы терминации транскрипции, такие как сигналы полиаденилирования и поли-U-последовательности). Такие регуляторные элементы описаны, например, в Goeddel, GENE EXPRESSION TECHNOLOGY: METHODS IN ENZYMOLOGY 185, Academic Press, San Diego, Calif. (1990). Регуляторные элементы включают такие, которые управляют конститутивной экспрессией нуклеотидной последовательности во многих типах клеток-хозяев, и такие, которые управляют экспрессией нуклеотидной последовательности только в определенных клетках-хозяевах (к примеру, тканеспецифичные регуляторные последовательности). Тканеспецифичный промотор может управлять экспрессией преимущественно в представляющей интерес целевой ткани, такой как мышца, нейрон, кость, кожа, кровь, конкретных органах (к примеру, печени, поджелудочной железе) или определенных типах клеток (к примеру, лимфоцитах). Регуляторные элементы также могут управлять экспрессией зависимым от времени образом, как, например, зависимым от клеточного цикла или зависимым от стадии развития образом, который может быть или может не быть также тканеспецифичным или специфичным к типу клеток. В некоторых вариантах осуществления вектор содержит один или несколько промоторов pol III (к примеру, 1, 2, 3, 4, 5 или более промоторов pol III), один или несколько промоторов pol II (к примеру, 1, 2, 3, 4, 5 или более промоторов pol II), один или несколько промоторов pol I (к примеру, 1, 2, 3, 4, 5 или более промоторов pol I) или их комбинации. Примеры промоторов pol III включают, без ограничения, промоторы U6 и H1. Примеры промоторов pol II включают, без ограничения, ретровирусный промотор LTR вируса саркомы Рауса (RSV) (необязательно с энхансером RSV), промотор цитомегаловируса (CMV) (необязательно с энхансером CMV) [см., например, Boshart et al., Cell, 41:521-530 (1985)], промотор SV40, промотор дигидрофолатредуктазы, промотор β-актина, промотор глицерофосфаткиназы (PGK) и промотор EF1α. Также выражением “регуляторный элемент” охвачены энхансерные элементы, такие как WPRE; энхансеры CMV; сегмент R-U5’ в LTR HTLV-I (Mol. Cell. Biol., Vol. 8(1), p. 466-472, 1988); энхансер SV40; и интронная последовательность между экзонами 2 и 3 β-глобина кролика (Proc. Natl. Acad. Sci. USA., Vol. 78(3), p. 1527-31, 1981). Специалистам в данной области будет понятно, что структура вектора экспрессии может зависеть от таких факторов, как выбор клетки хозяина, подлежащей трансформации, желательный уровень экспрессии и т.п. Вектор можно вводить в клетки-хозяева с получением, таким образом, транскриптов, белков или пептидов, в том числе слитых белков или пептидов, кодируемых нуклеиновыми кислотами, которые описаны в данном документе (к примеру, транскриптов коротких палиндромных повторов, регулярно расположенных группами (CRISPR), белков, ферментов, их мутантных форм, их слитых белков и т.п.).
Преимущественные векторы включают лентивирусы и аденоассоциированные вирусы, и типы таких векторов также могут быть выбраны для целенаправленного воздействия на определенные типы клеток.
В одном аспекте настоящее изобретение предусматривает эукариотическую клетку-хозяина, содержащую (a) первый регуляторный элемент, функционально связанный с парной tracr-последовательностью и одним или несколькими сайтами встраивания для встраивания одной или нескольких направляющих последовательностей выше парной tracr-последовательности, где при экспрессии направляющая последовательность управляет специфичным к последовательности связыванием комплекса CRISPR с целевой последовательностью в эукариотической клетке, где комплекс CRISPR содержит фермент CRISPR, образующий комплекс с (1) направляющей последовательностью, которая гибридизируется с целевой последовательностью, и (2) парной tracr-последовательностью, которая гибридизируется с tracr-последовательностью; и/или (b) второй регуляторный элемент, функционально связанный с кодирующей фермент последовательностью, кодирующей указанный фермент CRISPR, содержащий последовательность ядерной локализации. В некоторых вариантах осуществления клетка-хозяин содержит компоненты (a) и (b). В некоторых вариантах осуществления компонент (a), компонент (b) или компоненты (a) и (b) стабильно интегрируются в геном эукариотической клетки-хозяина. В некоторых вариантах осуществления компонент (a) дополнительно содержит tracr-последовательность ниже парной tracr-последовательности под контролем первого регуляторного элемента. В некоторых вариантах осуществления компонент (a) дополнительно содержит две или более направляющие последовательности, функционально связанные с первым регуляторным элементом, где при экспрессии каждая из двух или более направляющих последовательностей управляет специфичным к последовательности связыванием комплекса CRISPR со своей целевой последовательностью в эукариотической клетке. В некоторых вариантах осуществления эукариотическая клетка-хозяин дополнительно содержит третий регуляторный элемент, такой как промотор полимеразы III, функционально связанный с указанной tracr-последовательностью. В некоторых вариантах осуществления tracr-последовательность характеризуется по меньшей мере 50%, 60%, 70%, 80%, 90%, 95% или 99% комплементарности последовательности по длине парной tracr-последовательности при оптимальном выравнивании. Фермент может быть гомологом или ортологом Cas9. В некоторых вариантах осуществления фермент CRISPR кодон-оптимизирован для экспрессии в эукариотической клетке. В некоторых вариантах осуществления фермент CRISPR управляет расщеплением одной или двух нитей в определенной точке целевой последовательности. В некоторых вариантах осуществления у фермента CRISPR отсутствует активность для расщепления нитей ДНК. В некоторых вариантах осуществления первый регуляторный элемент является промотором полимеразы III. В некоторых вариантах осуществления второй регуляторный элемент является промотором полимеразы II. В некоторых вариантах осуществления направляющая последовательность составляет по меньшей мере 15, 16, 17, 18, 19, 20, 25 нуклеотидов, или от 10 до 30, или от 15 до 25, или от 15 до 20 нуклеотидов в длину. В одном аспекте настоящее изобретение предусматривает отличный от человека эукариотический организм, предпочтительно многоклеточный эукариотический организм, содержащий эукариотическую клетку-хозяина согласно любому из описанных вариантов осуществления. В других аспектах настоящее изобретение предусматривает эукариотический организм, предпочтительно многоклеточный эукариотический организм, содержащий эукариотическую клетку-хозяина согласно любому из описанных вариантов осуществления. Организм в некоторых вариантах осуществления данных аспектов может быть животным, например, млекопитающим. Также организмом может быть членистоногое, как, например, насекомое. Организмом также может быть растение. Кроме того, организмом может быть гриб.
В одном аспекте настоящее изобретение предусматривает набор, содержащий один или несколько компонентов, описанных в данном документе. В некоторых вариантах осуществления набор содержит векторную систему и инструкции по применению набора. В некоторых вариантах осуществления векторная система содержит (a) первый регуляторный элемент, функционально связанный с парной tracr-последовательностью и одним или несколькими сайтами встраивания для встраивания одной или нескольких направляющих последовательностей выше парной tracr-последовательности, где при экспрессии направляющая последовательность управляет специфичным к последовательности связыванием комплекса CRISPR с целевой последовательностью в эукариотической клетке, где комплекс CRISPR содержит фермент CRISPR, образующий комплекс с (1) направляющей последовательностью, которая гибридизируется с целевой последовательностью, и (2) парной tracr-последовательностью, которая гибридизируется с tracr-последовательностью; и/или (b) второй регуляторный элемент, функционально связанный с кодирующей фермент последовательностью, кодирующей указанный фермент CRISPR, содержащий последовательность ядерной локализации. В некоторых вариантах осуществления набор содержит компоненты (a) и (b), находящиеся в одном и том же или в разных векторах системы. В некоторых вариантах осуществления компонент (a) дополнительно содержит tracr-последовательность ниже парной tracr-последовательности под контролем первого регуляторного элемента. В некоторых вариантах осуществления компонент (a) дополнительно содержит две или более направляющие последовательности, функционально связанные с первым регуляторным элементом, где при экспрессии каждая из двух или более направляющих последовательностей управляет специфичным к последовательности связыванием комплекса CRISPR со своей целевой последовательностью в эукариотической клетке. В некоторых вариантах осуществления система дополнительно содержит третий регуляторный элемент, такой как промотор полимеразы III, функционально связанный с указанной tracr-последовательностью. В некоторых вариантах осуществления tracr-последовательность характеризуется по меньшей мере 50%, 60%, 70%, 80%, 90%, 95% или 99% комплементарности последовательности по длине парной tracr-последовательности при оптимальном выравнивании. В некоторых вариантах осуществления фермент CRISPR содержит одну или несколько последовательностей ядерной локализации, достаточно эффективных, чтобы управлять накоплением указанного фермента CRISPR в обнаруживаемом количестве в ядре эукариотической клетки. В некоторых вариантах осуществления фермент CRISPR является ферментом системы CRISPR II типа. В некоторых вариантах осуществления фермент CRISPR является ферментом Cas9. В некоторых вариантах осуществления фермент Cas9 является Cas9 S. pneumoniae, S. pyogenes или S. thermophilus и может включать мутированный Cas9, полученный из этих организмов. Фермент может быть гомологом или ортологом Cas9. В некоторых вариантах осуществления фермент CRISPR кодон-оптимизирован для экспрессии в эукариотической клетке. В некоторых вариантах осуществления фермент CRISPR управляет расщеплением одной или двух нитей в определенной точке целевой последовательности. В некоторых вариантах осуществления у фермента CRISPR отсутствует активность для расщепления нитей ДНК. В некоторых вариантах осуществления первый регуляторный элемент является промотором полимеразы III. В некоторых вариантах осуществления второй регуляторный элемент является промотором полимеразы II. В некоторых вариантах осуществления направляющая последовательность составляет по меньшей мере 15, 16, 17, 18, 19, 20, 25 нуклеотидов, или от 10 до 30, или от 15 до 25, или от 15 до 20 нуклеотидов в длину.
В одном аспекте настоящее изобретение предусматривает способ модификации целевого полинуклеотида в эукариотической клетке. В некоторых вариантах осуществления способ включает обеспечение связывания комплекса CRISPR с целевым полинуклеотидом для осуществления расщепления указанного целевого полинуклеотида с модификацией, таким образом, целевого полинуклеотида, где комплекс CRISPR содержит фермент CRISPR, образующий комплекс с направляющей последовательностью, гибридизирующейся с целевой последовательностью в указанном целевом полинуклеотиде, где указанная направляющая последовательность связана с парной tracr-последовательностью, которая, в свою очередь, гибридизируется с tracr-последовательностью. В некоторых вариантах осуществления указанное расщепление включает расщепление одной или двух нитей в определенной точке целевой последовательности указанным ферментом CRISPR. В некоторых вариантах осуществления указанное расщепление приводит к сниженной транскрипции целевого гена. В некоторых вариантах осуществления способ дополнительно включает репарацию указанного расщепленного целевого полинуклеотида при помощи гомологичной рекомбинации с экзогенным матричным полинуклеотидом, где указанная репарация приводит к мутации, включающей вставку, делецию или замену одного или нескольких нуклеотидов указанного целевого полинуклеотида. В некоторых вариантах осуществления указанная мутация приводит к одной или нескольким аминокислотным заменам в белке, экспрессируемом с гена, содержащего целевую последовательность. В некоторых вариантах осуществления способ дополнительно включает доставку одного или нескольких векторов в указанную эукариотическую клетку, где один или несколько векторов управляют экспрессией одного или нескольких из: фермента CRISPR, направляющей последовательности, связанной с парной tracr-последовательностью, и tracr-последовательности. В некоторых вариантах осуществления указанные векторы доставляют в эукариотическую клетку в субъекте. В некоторых вариантах осуществления указанная модификация имеет место в указанной эукариотической клетке в клеточной культуре. В некоторых вариантах осуществления способ дополнительно включает выделение указанной эукариотической клетки из субъекта перед указанной модификацией. В некоторых вариантах осуществления способ дополнительно включает возвращение указанной эукариотической клетки и/или клеток, полученных из субъекта, указанному субъекту.
В одном аспекте настоящее изобретение предусматривает способ модификации экспрессии полинуклеотида в эукариотической клетке. В некоторых вариантах осуществления способ включает обеспечение связывания комплекса CRISPR с полинуклеотидом так, что указанное связывание приводит к повышенной или пониженной экспрессии указанного полинуклеотида; где комплекс CRISPR содержит фермент CRISPR, образующий комплекс с направляющей последовательностью, гибридизирующейся с целевой последовательностью в указанном целевом полинуклеотиде, где указанная направляющая последовательность связана с парной tracr-последовательностью, которая, в свою очередь, гибридизируется с tracr-последовательностью. В некоторых вариантах осуществления способ дополнительно включает доставку одного или нескольких векторов в указанные эукариотические клетки, где один или несколько векторов управляют экспрессией одного или нескольких из: фермента CRISPR, направляющей последовательности, связанной с парной tracr-последовательностью, и tracr-последовательности.
В одном аспекте настоящее изобретение предусматривает способ получения модельной эукариотической клетки, содержащей мутированный ген, ответственный за развитие заболевания. В некоторых вариантах осуществления ген, ответственный за развитие заболевания, представляет собой любой ген, ассоциированный с повышением риска наличия или развития заболевания. В некоторых вариантах осуществления способ включает (a) введение одного или нескольких векторов в эукариотическую клетку, где один или несколько векторов управляют экспрессией одного или нескольких из: фермента CRISPR, направляющей последовательности, связанной с парной tracr-последовательностью, и tracr-последовательности; и (b) обеспечение связывания комплекса CRISPR с целевым полинуклеотидом для осуществления расщепления целевого полинуклеотида в указанном гене, ответственном за развитие заболевания, где комплекс CRISPR содержит фермент CRISPR, образующий комплекс с (1) направляющей последовательностью, которая гибридизируется с целевой последовательностью в целевом полинуклеотиде, и (2) парной tracr-последовательностью, которая гибридизируется с tracr-последовательностью, таким образом, получая модельную эукариотическую клетку, содержащую мутированный ген, ответственный за развитие заболевания. В некоторых вариантах осуществления указанное расщепление включает расщепление одной или двух нитей в определенной точке целевой последовательности указанным ферментом CRISPR. В некоторых вариантах осуществления указанное расщепление приводит к сниженной транскрипции целевого гена. В некоторых вариантах осуществления способ дополнительно включает репарацию указанного расщепленного целевого полинуклеотида при помощи гомологичной рекомбинации с экзогенным матричным полинуклеотидом, где указанная репарация приводит к мутации, включающей вставку, делецию или замену одного или нескольких нуклеотидов указанного целевого полинуклеотида. В некоторых вариантах осуществления указанная мутация приводит к одной или нескольким аминокислотным заменам при экспрессии белка с гена, содержащего целевую последовательность.
В одном аспекте настоящее изобретение предусматривает способ получения биологически активного средства, которое модулирует процесс передачи сигнала в клетке, ассоциированный с геном, ответственным за развитие заболевания. В некоторых вариантах осуществления ген, ответственный за развитие заболевания, представляет собой любой ген, ассоциированный с повышением риска наличия или развития заболевания. В некоторых вариантах осуществления способ включает (a) приведение тестового соединения в контакт с модельной клеткой по любому одному из описанных вариантов осуществления и (b) обнаружение изменения при считывании, которое свидетельствует об уменьшении или усилении процесса передачи сигнала в клетке, ассоциированного с указанной мутацией в указанном гене, ответственном за развитие заболевания, с получением, таким образом, указанного биологически активного средства, которое модулирует указанный процесс передачи сигнала в клетке, ассоциированный с указанным геном, ответственным за развитие заболевания.
В одном аспекте настоящее изобретение предусматривает рекомбинантный полинуклеотид, содержащий направляющую последовательность выше парной tracr-последовательности, где направляющая последовательность при экспрессии управляет специфичным к последовательности связыванием комплекса CRISPR с соответствующей целевой последовательностью, присутствующей в эукариотической клетке. В некоторых вариантах осуществления целевая последовательность является вирусной последовательностью, присутствующей в эукариотической клетке. В некоторых вариантах осуществления целевая последовательность является протоонкогеном или онкогеном.
В одном аспекте настоящее изобретение предусматривает способ отбора одной или нескольких клеток путем введения одной или нескольких мутаций в ген в одной или нескольких клетках, при этом способ включает введение одного или нескольких векторов в клетку(и), где один или несколько векторов управляют экспрессией одного или нескольких из: фермента CRISPR, направляющей последовательности, связанной с парной tracr-последовательностью, tracr-последовательности и матрицы редактирования; где матрица редактирования содержит одну или несколько мутаций, которые прекращают расщепление фермента CRISPR; обеспечение гомологичной рекомбинации матрицы редактирования с целевым полинуклеотидом в отбираемой(ых) клетке(ах); обеспечение связывания комплекса CRISPR с целевым полинуклеотидом для осуществления расщепления целевого полинуклеотида в указанном гене, где комплекс CRISPR содержит фермент CRISPR, образующий комплекс с (1) направляющей последовательностью, которая гибридизируется с целевой последовательностью в целевом полинуклеотиде, и (2) парной tracr-последовательностью, которая гибридизируется с tracr-последовательностью, где связывание комплекса CRISPR с целевым полинуклеотидом индуцирует гибель клеток, с обеспечением тем самым отбора одной или нескольких клеток, в которые были введены одна или несколько мутаций. В некоторых вариантах осуществления фермент CRISPR является ферментом системы CRISPR II типа. В некоторых вариантах осуществления фермент CRISPR представляет собой белок Cas9. В некоторых вариантах осуществления белок Cas9 является Cas9 S. pneumoniae, S. pyogenes или S. thermophilus и может включать мутированный Cas9, полученный из этих организмов. Фермент может быть гомологом или ортологом Cas9. В некоторых вариантах осуществления фермент кодон-оптимизирован для экспрессии в эукариотической клетке. В некоторых вариантах осуществления фермент управляет расщеплением одной или двух нитей в определенной точке целевой последовательности. В предпочтительном варианте осуществления фермент CRISPR представляет собой Cas9. В другом предпочтительном варианте настоящего изобретения отбираемая клетка может быть эукариотической клеткой. Аспекты настоящего изобретения обеспечивают возможность отбора конкретных клеток без необходимости наличия маркера отбора или двухстадийного способа, который может включать систему негативного отбора.
Аспекты настоящего изобретения охватывают сайт-специфический нокаут гена в эндогенном геноме. Настоящее изобретение является более преимущественным по сравнению с применением технологий с использованием сайт-специфических нуклеаз, базовыми элементами которых являются "цинковые пальцы" и TAL-эффекторы, поскольку оно не требует специальной разработки и может быть применено для одновременного нокаута нескольких генов в одном геноме. В дополнительном аспекте настоящее изобретение охватывает сайт-специфическое редактирование генома. Настоящее изобретение является более преимущественным по сравнению с применением природных или искусственных сайт-специфических нуклеаз или рекомбиназ, поскольку с помощью него может быть возможным введение сайт-специфических двухцепочечных разрывов для обеспечения гомологичной рекомбинации в целевых геномных локусах. В другом аспекте настоящее изобретение охватывает специфичную к последовательности ДНК-интерференцию. Настоящее изобретение можно применять для инактивации генома вредоносных организмов, имеющих ДНК, таких как микроорганизмы, вирусы или даже раковые клетки, путем непосредственного введения разрывов в определенные сайты в геноме этих организмов. Настоящее изобретение предусматривает способы и композиции для мультиплексного конструирования генома, поскольку система CRISPR-Cas по настоящему изобретению может быть без труда нацелена на множество сайтов в геноме посредством применения множества специфичных к последовательности спейсерных элементов или направляющих последовательностей CRISPR.
Соответственно, целью настоящего изобретения не является охват в пределах настоящего изобретения любого ранее известного продукта, способа получения продукта или способа применения продукта, так что заявители оставляют за собой право и настоящим раскрывают отказ от прав на любой ранее известный продукт, процесс или способ. Следует дополнительно отметить, что настоящее изобретение не предназначено охватывать в пределах объема настоящего изобретения любой продукт, способ получения продукта или способ применения продукта, который не соответствует письменному описанию и требованиям достаточного раскрытия сути изобретения USPTO (первый пункт § 112 статьи 35 USC) или EPO (статья 83 EPC), так что заявители оставляют за собой право и настоящим раскрывают отказ от прав на любой ранее описанный продукт, способ получения продукта или способ применения продукта.
Следует отметить, что в данном раскрытии и особенно в формуле изобретения и/или параграфах такие выражения, как "содержит", "содержащийся", "содержащий" и т.п., могут иметь значение, приписываемое им в патентном законодательстве США, например, они могут означать "включает", "включенный", "включающий" и т.п., и что такие выражения, как "состоящий, по сути, из" и "состоит, по сути, из" имеют значение, приписываемое им в патентном законодательстве США, например, они допускают не указанные прямо элементы, но исключают элементы, которые имеются в известном уровне техники или которые влияют на основные или новые характеристики настоящего изобретения. Эти и другие варианты осуществления раскрыты или являются очевидными, исходя из следующего подробного описания, и охвачены им.
Краткое описание графических материалов
Новые признаки настоящего изобретения изложены с характерными особенностями в прилагаемой формуле изобретения. Лучшее понимание признаков и преимуществ настоящего изобретения будет доступно благодаря ссылке на следующее подробное описание, в котором изложены показательные варианты осуществления, в которых используют принципы настоящего изобретения, и на сопутствующие графические материалы.
На фигуре 1 изображена схематическая модель системы CRISPR. Нуклеаза Cas9 из Streptococcus pyogenes (желтый) нацелена на геномную ДНК при помощи синтетической направляющей РНК (sgRNA), состоящей из 20-нуклеотидной направляющей последовательности (голубой) и каркаса (красный). Направляющая последовательность образует пары оснований с ДНК-мишенью (голубой) непосредственно выше необходимого мотива, смежного с протоспейсером (PAM; пурпурный), 5’-NGG, и Cas9 опосредует двухцепочечный разрыв (DSB) на ~3 п.о. выше PAM (красный треугольник).
На фигурах 2A-F показана иллюстративная система CRISPR, возможный механизм действия, пример адаптации для экспрессии в эукариотических клетках и результаты тестов, оценивающих ядерную локализацию и активность CRISPR. На фигуре 2C раскрываются SEQ ID NO 23-24, соответственно, в порядке встречаемости. На фигуре 2E раскрываются SEQ ID NO 25-27, соответственно, в порядке встречаемости. На фигуре 2F раскрываются SEQ ID NO 28-32, соответственно, в порядке встречаемости.
На фигуре 3A-D показаны результаты оценки специфичности SpCas9 в отношении мишени-примера. На фигуре 3А раскрываются SEQ ID NO 34-44, соответственно, в порядке встречаемости. На фигуре 3C раскрывается SEQ ID NO: 33.
На фигурах 4A-G показана иллюстративная векторная система и результаты ее применения при управлении гомологичной рекомбинацией в эукариотических клетках. На фигуре 4E раскрывается SEQ ID NO: 45. На фигуре 4F раскрываются SEQ ID NO 46-47, соответственно, в порядке встречаемости. На фигуре 4G раскрываются SEQ ID NO 48-52, соответственно, в порядке встречаемости.
На фигуре 5 представлена таблица последовательностей протоспейсеров (SEQ ID NO 16, 15, 14, 53-58, 18, 17 и 59-63, соответственно, в порядке встречаемости) и обобщены результаты касательно эффективности модификаций для протоспейсеров-мишеней, разработанных на основе иллюстративных систем CRISPR S. pyogenes и S. thermophilus с соответствующими PAM, взаимодействующими с локусами в геномах человека и мыши. Клетки трансфицировали Cas9 и либо pre-crRNA/tracrRNA, либо химерной РНК и анализировали через 72 часа после трансфекции. Процент вставок/делеций рассчитывали на основе результатов анализа с помощью Surveyor с указанными линиями клеток (N=3 для всех протоспейсеров-мишеней, ошибки представляют собой стандартные ошибки среднего, "N.D." означает "не обнаруживается посредством анализа с помощью Surveyor", и "N.T." означает "не тестировали в данном исследовании").
На фигурах 6A-C показано сравнение различных транскриптов tracrRNA для опосредованного Cas9 целенаправленного воздействия на ген. На фигуре 6А раскрываются SEQ ID NO 64-65, соответственно, в порядке встречаемости.
На фигуре 7 показано схематическое изображение анализа с помощью нуклеазы Surveyor для обнаружения индуцированных двухцепочечным разрывом микровставок и микроделеций.
На фигурах 8A-B показаны иллюстративные бицистронные векторы экспрессии для экспрессии элементов системы CRISPR в эукариотических клетках. На фигуре 8А раскрываются SEQ ID NO 66-68, соответственно, в порядке встречаемости. На фигуре 8В раскрываются SEQ ID NO 69-71, соответственно, в порядке встречаемости.
На фигурах 9A-C показаны гистогораммы расстояний между смежными PAM (NGG) локуса 1 S. pyogenes SF370 (фигура 9A) и PAM (NNAGAAW) локуса 2 LMD9 S. thermophilus (фигура 9B) в геноме человека и расстояния для каждого PAM в хромосомах (Chr) (фигура 9C).
На фигурах 10A-D показана иллюстративная система CRISPR, пример адаптации для экспрессии в эукариотических клетках и результаты тестов, оценивающих активность CRISPR. На фигуре 10В раскрываются SEQ ID NO 72-73, соответственно, в порядке встречаемости. На фигуре 10C раскрывается SEQ ID NO: 74.
На фигурах 11A-C показаны иллюстративные манипуляции с системой CRISPR для целенаправленного воздействия на геномные локусы в клетках млекопитающего. На фигуре 11А раскрывается SEQ ID NO: 75. На фигуре 11В раскрываются SEQ ID NO 76-78, соответственно, в порядке встречаемости.
На фигурах 12A-B показаны результаты анализа нозерн-блоттинга процессинга crRNA в клетках млекопитающего. На фигуре 12А раскрывается SEQ ID NO: 79.
На фигурах 13A-B показан иллюстративный отбор протоспейсеров в локусах PVALB человека и Th мыши. На фигуре 13А раскрывается SEQ ID NO: 80. На фигуре 13В раскрывается SEQ ID NO: 81.
На фигуре 14 показан иллюстративный протоспейсер и соответствующие последовательности PAM-мишени системы CRISPR S. thermophilus в локусе EMX1 человека. На фигуре 14 раскрывается SEQ ID NO: 74.
На фигуре 15 представлена таблица последовательностей (SEQ ID NO 82-93, соответственно, в порядке встречаемости) для праймеров и зондов, используемых для Surveyor, RFLP, геномного секвенирования и анализов нозерн-блоттинга.
На фигурах 16A-C показана иллюстративная манипуляция с системой CRISPR с химерными РНК и результаты анализов с помощью SURVEYOR в отношении активности системы в эукариотических клетках. На фигуре 16А раскрывается SEQ ID NO: 94.
На фигурах 17A-B показано графическое изображение результатов анализа с помощью SURVEYOR в отношении активности системы CRISPR в эукариотических клетках.
На фигуре 18 показано иллюстративное отображение некоторых целевых сайтов для Cas9 S. pyogenes в геноме человека, полученное с использованием геномного браузера UCSC. На фигуре 18 раскрываются SEQ ID NO 95-173, соответственно, в порядке встречаемости.
На фигурах 19A-D показано круговое отображение филогенетического анализа, выявляющего пять семейств Cas9, включая три группы больших Cas9 (~1400 аминокислот) и две малых Cas9 (~1100 аминокислот).
На фигурах 20A-F показано линейное отображение филогенетического анализа, выявляющего пять семейств Cas9, включая три группы больших Cas9 (~1400 аминокислоты) и две малых Cas9 (~1100 аминокислоты).
На фигурах 21A-D показано редактирование генома посредством гомологичной рекомбинации. (a) Схематическое изображение никазы SpCas9 с мутацией D10A в каталитическом домене RuvC I. (b) Схематическое представление гомологичной рекомбинации (HR) в локусе EMX1 человека при использовании смысловых или антисмысловых однонитевых олигонуклеотидов в качестве матриц для репарации. Красная стрелка вверху указывает на сайт расщепления для sgRNA; ПЦР праймеры для для генотипирования (таблицы J и K) обозначены стрелками в правой секции. На фигуре 21C раскрываются SEQ ID NO 174-176, 174, 177 и 176, соответственно, в порядке встречаемости. (c) Последовательность участка, модифицированного с помощью HR. (d) Анализ вставок/делеций в целевом локусе 1 EMX1 (n = 3), опосредованных SpCas9 дикого типа (wt) и никазой SpCas9 (D10A), с помощью SURVEYOR. Стрелки указывают положения фрагментов ожидаемого размера.
На фигурах 22A-B показаны одиночные векторные структуры для SpCas9. На фигуре 22А раскрываются SEQ ID NO 178-180, соответственно, в порядке встречаемости. На фигуре 22В раскрывается SEQ ID NO: 181.
Фигуры приведены в данном документе только в целях иллюстрации, и они не обязательно изображены в масштабе.
Подробное описание изобретения
Выражения “полинуклеотид”, “нуклеотид”, “нуклеотидная последовательность”, “нуклеиновая кислота” и “олигонуклеотид” используют взаимозаменяемо. Они обозначают полимерную форму нуклеотидов любой длины, как дезоксирибонуклеотидов, так и рибонуклеотидов или их аналогов. Полинуклеотиды могут обладать любой пространственной структурой и могут выполнять любую функцию, известную или не известную. Неограничивающими примерами полинуклеотидов являются следующие: кодирующие или некодирующе участки гена или фрагмента гена, локусы(локус), определенные в результате анализа сцепления, экзоны, интроны, матричная РНК (мРНК), транспортная РНК, рибосомная РНК, короткая интерферирующая РНК (siRNA), короткая шпилечная РНК (shRNA), микроРНК (miRNA), рибозимы, кДНК, рекомбинантные полинуклеотиды, разветвленные полинуклеотиды, плазмиды, векторы, выделенная ДНК любой последовательности, выделенная РНК любой последовательности, зонды для нуклеиновых кислот и праймеры. Полинуклеотид может содержать один или несколько модифицированных нуклеотидов, как, например, метилированные нуклеотиды и аналоги нуклеотидов. При наличии, модификации в нуклеотидную структуру могут быть внесены до или после сборки полимера. Последовательность нуклеотидов может прерываться отличными от нуклеотидов компонентами. Полинуклеотид можно дополнительно модифицировать после полимеризации, как, например, путем конъюгации с компонентом для мечения.
В аспектах настоящего изобретения выражения “химерная РНК”, “химерная направляющая РНК”, “направляющая РНК”, “одиночная направляющая РНК” и “синтетическая направляющая РНК” используют взаимозаменяемо, и они обозначают полинуклеотидную последовательность, содержащую направляющую последовательность, tracr-последовательность и парную tracr-последовательность. Выражение “направляющая последовательность” обозначает последовательность из приблизительно 20 п.о. в пределах направляющей РНК, которая определяет целевой сайт, и его можно использовать взаимозаменяемо с выражениями “гид” или “спейсер”. Выражение “парная tracr-последовательность” также можно использовать взаимозаменяемо с выражением “прямой(ые) повтор(ы)”. Иллюстративная система CRISPR-Cas изображена на фигуре 1.
Используемое в данном документе выражение “дикий тип” является выражением из данной области, понятным специалисту в данной области, и означает типичную форму организма, штамма, гена или характеристики, которые встречаются в природе в отличие от мутантных или вариантных форм.
Используемое в данном документе выражение “вариант” следует понимать как означающее проявление качеств, которые характеризуются паттерном, который отличается от такового, встречающегося в природе.
Выражения “не встречающийся в природе” или “сконструированный” используют взаимозаменяемо, и они указывают на вмешательство человека. Выражения, в тех случаях, когда они касаются молекул нуклеиновых кислот или полипептидов, означают, что молекула нуклеиновой кислоты или полипептид по меньшей мере практически не содержат по меньшей мере один отличный компонент, с которым они естественным образом связаны в природе и встречаются в природе.
“Комплементарность” означает способность нуклеиновой кислоты образовывать водородную(ые) связь(и) с другой последовательностью нуклеиновой кислоты при помощи либо традиционного спаривания оснований по Уотсону-Крику, либо других нетрадиционных типов. Процент комплементарности показывает процентную долю остатков в молекуле нуклеиновой кислоты, которые могут образовывать водородные связи (к примеру, спаривание оснований по Уотсону-Крику) со второй последовательностью нуклеиновой кислоты (к примеру, при этом 5, 6, 7, 8, 9, 10 из 10 будут на 50%, 60%, 70%, 80%, 90% и 100% комплементарны). “Точная комплементарность” означает, что все смежные остатки последовательности нуклеиновой кислоты будут связаны водородными связями с тем же количеством смежных остатков во второй последовательности нуклеиновой кислоты. Выражение “практически комплементарный”, используемое в данном документе, означает степень комплементарности, которая составляет по меньшей мере 60%, 65%, 70%, 75%, 80%, 85%, 90%, 95%, 97%, 98%, 99% или 100% в пределах участка из 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 30, 35, 40, 45, 50 или более нуклеотидов, или относится к двум нуклеиновым кислотам, которые гибиридизируются при жестких условиях.
Используемые в данном документе “жесткие условия” в отношении гибридизации означают условия, при которых нуклеиновая кислота с комплементарностью к целевой последовательности преимущественно гибридизируется с целевой последовательностью и практически не гибридизируется с нецелевыми последовательностями. Жесткие условия, как правило, являются зависимыми от последовательности и изменяются в зависимости от ряда факторов. Как правило, чем длиннее последовательность, тем выше температура, при которой последовательность специфично гибридизируется с целевой последовательностью. Неограничивающие примеры жестких условий описаны подробно в Tijssen (1993), Laboratory Techniques In Biochemistry And Molecular Biology-Hybridization With Nucleic Acid Probes Part I, Second Chapter “Overview of principles of hybridization and the strategy of nucleic acid probe assay”, Elsevier, N.Y.
“Гибридизация” означает реакцию, при которой один или несколько полинуклеотидов реагируют с образованием комплекса, который стабилизируется посредством образования водородных связей между основаниями нуклеотидных остатков. Образование водородных связей может происходить по принципу спаривания оснований по Уотсону-Крику, Хугстиновского связывания или любым другим специфичным к последовательности образом. Комплекс может содержать две нити, образующие дуплексную структуру, три или более нитей, образующих многонитевой комплекс, одиночную самогибридизирующуюся нить или любую их комбинацию. Реакция гибридизации может представлять собой стадию в более обширном способе, такую как начальная стадия ПЦР или расщепление полинуклеотида при помощи фермента. Последовательность, способную гибридизироваться с данной последовательностью, называют “комплементарной последовательностью” данной последовательности.
Используемое в данном документе выражение “экспрессия” означает процесс, при котором полинуклеотид транскрибируется с ДНК-матрицы (как, например, с образованием мРНК или другого РНК-транскрипта), и/или способ, при помощи которого транскрибированная мРНК далее транслируется с образованием пептидов, полипептидов или белков. Транскрипты и кодируемые полипептиды можно в совокупности называть “продукт гена”. Если полинуклеотид получен из геномной ДНК, то экспрессия может включать сплайсинг мРНК в эукариотической клетке.
Выражения “полипептид”, “пептид” и “белок” используют взаимозаменяемо в данном документе для обозначения полимеров из аминокислот любой длины. Полимер может быть линейным или разветвленным, он может содержать модифицированные аминокислоты, и его структура может прерываться отличными от аминокислот компонентами. Выражения также охватывают полимер из аминокислот, который был модифицирован; например, образованием дисульфидных связей, гликозилированием, липидизацией, ацетилированием, фосфорилированием или любой другой манипуляцией, такой как соединение с компонентом для мечения. Используемое в данном документе выражение “аминокислота” включает природные, и/или отличные от природных, или синтетические аминокислоты, в том числе глицин и как D-, так и L-оптические изомеры, и аналоги аминокислот, и пептидомиметики.
Выражения “субъект”, “индивидуум” и “пациент” используют взаимозаменяемо в данном документе для обозначения позвоночного, предпочтительно млекопитающего, более предпочтительно человека. Млекопитающие включают, без ограничения, мышей, обезьян, людей, сельскохозяйственных животных, животных для спорта и домашних животных. Также охватываются ткани, клетки и их потомство биологического организма, полученные in vivo или культивированные in vitro.
Выражения “терапевтическое средство”, “оказывающее терапевтический эффект средство” или “средство для лечения” используют взаимозаменяемо, и они означают молекулу или соединение, которые оказывают некоторое благоприятное воздействие при введении субъекту. Благоприятное воздействие включает осуществление диагностических определений; облегчение заболевания, симптома, нарушения или патологического состояния; ослабление или предупреждение начала проявления заболевания, симптома, нарушения или состояния; а также общее противодействие заболеванию, симптому, нарушению или патологическому состоянию.
Используемые в данном документе выражения “лечение”, или “осуществление лечения”, или “временное ослабление”, или “облегчение” используют взаимозаменяемо. Эти выражения означают подход для получения благоприятных или желательных результатов, в том числе, без ограничения, терапевтического эффекта и/или профилактического эффекта. Под терапевтическим эффектом понимают любые терапевтически значимые улучшение или действие в отношении одного или нескольких заболеваний, состояний или симптомов при лечении. Для профилактического эффекта композиции можно вводить субъекту с риском развития конкретного заболевания, состояния или симптома или субъекту, который сообщает об одном или нескольких физиологических симптомах заболевания, даже если заболевание, состояние или симптом могли еще не проявиться.
Выражение “эффективное количество” или “терапевтически эффективное количество” означает количество средства, которого достаточно для обеспечения благоприятных или желательных результатов. Терапевтически эффективное количество может изменяться в зависимости от одного или нескольких из: субъекта и болезненного состояния, которые подлежат лечению, веса и возраста субъекта, тяжести болезненного состояния, способа введения и подобного, что специалист в данной области легко может определить. Выражение также применимо к дозе, с помощью которой можно получить изображение для определения любым одним из способов визуализации, описанных в данном документе. Конкретная доза может изменяться в зависимости от одного или нескольких из: конкретного выбранного средства, режима дозирования, которому следуют, того, вводят ли его в комбинации с другими средствами, выбора времени введения, визуализируемой ткани и физической системы доставки, в которой оно заключено.
Практическое применение настоящего изобретения предусматривает, если не указано иное, традиционные методики иммунологии, биохимии, химии, молекулярной биологии, микробиологии, клеточной биологии, геномики и технологию рекомбинантной ДНК, которые находятся в пределах квалификации специалиста в данной области. См. Sambrook, Fritsch and Maniatis, MOLECULAR CLONING: A LABORATORY MANUAL, 2nd edition (1989); CURRENT PROTOCOLS IN MOLECULAR BIOLOGY (F. M. Ausubel, et al. eds., (1987)); серия METHODS IN ENZYMOLOGY (Academic Press, Inc.): PCR 2: A PRACTICAL APPROACH (M.J. MacPherson, B.D. Hames and G.R. Taylor eds. (1995)), Harlow and Lane, eds. (1988) ANTIBODIES, A LABORATORY MANUAL и ANIMAL CELL CULTURE (R.I. Freshney, ed. (1987)).
Некоторые аспекты настоящего изобретения касаются векторных систем, содержащих один или несколько векторов, или векторов как таковых. Векторы могут быть разработаны для экспрессии транскриптов CRISPR (к примеру, транскриптов нуклеиновых кислот, белков или ферментов) в прокариотических или эукариотических клетках. Например, транскрипты CRISPR могут экспрессироваться в бактериальных клетках, как, например, Escherichia coli, клетках насекомых (с использованием бакуловирусных векторов экспрессии), клетках дрожжей или клетках млекопитающих. Подходящие клетки-хозяева дополнительно рассматриваются в Goeddel, GENE EXPRESSION TECHNOLOGY: METHODS IN ENZYMOLOGY 185, Academic Press, San Diego, Calif. (1990). В качестве альтернативы, рекомбинантный вектор экспрессии может транскрибироваться и транслироваться in vitro, например, при помощи регуляторных последовательностей промотора T7 и полимеразы T7.
Векторы можно вводить и размножать в прокариоте. В некоторых вариантах осуществления прокариота используют для амплификации копий вектора, который предполагается вводить в эукариотическую клетку, или в качестве промежуточного вектора при получении вектора, который предполагается вводить в эукариотическую клетку (к примеру, путем амплификации плазмиды как части системы упаковки вирусного вектора). В некоторых вариантах осуществления прокариота используют для амплификации копий вектора и экспрессии одной или нескольких нуклеиновых кислот, как, например, для обеспечения источника одного или нескольких белков для доставки в клетку-хозяина или организм-хозяин. Экспрессию белков в прокариотах наиболее часто осуществляют в Escherichia coli с векторами, содержащими конститутивные или индуцибельные промоторы, управляющие экспрессией либо слитых белков, либо отличных от слитых белков. Слитые векторы добавляют некоторое количество аминокислот к белку, закодированному в них, как, например, к амино-концу рекомбинантного белка. Такие слитые векторы могут служить для одной или нескольких целей, как например: (i) для повышения экспрессии рекомбинантного белка; (ii) для повышения растворимости рекомбинантного белка и (iii) для содействия очистке рекомбинантного белка посредством функционирования в качестве лиганда при аффинной очистке. Часто в слитые векторы экспрессии вводят сайт протеолитического расщепления в месте соединения фрагмента слияния и рекомбинантного белка для облегчения отделения рекомбинантного белка от фрагмента слияния после очистки слитого белка. Такие ферменты и их когнатные распознающие последовательности включают фактор Xa, тромбин и энтерокиназу. Иллюстративные слитые векторы экспрессии включают pGEX (Pharmacia Biotech Inc; Smith and Johnson, 1988. Gene 67: 31-40), pMAL (New England Biolabs, Беверли, Массачусетс) и pRIT5 (Pharmacia, Пискатауэй, Нью-Джерси), в которых глутатион-S-трансфераза (GST), мальтоза-связывающий белок E или белок A, соответственно, слиты с целевым рекомбинантным белком.
Примеры подходящих индуцибельных не являющихся слитыми векторов экспрессии E. coli включают pTrc (Amrann et al., (1988) Gene 69:301-315) и pET 11d (Studier et al., GENE EXPRESSION TECHNOLOGY: METHODS IN ENZYMOLOGY 185, Academic Press, San Diego, Calif. (1990) 60-89).
В некоторых вариантах осуществления вектор является вектором экспрессии для дрожжей. Примеры векторов для экспрессии в дрожжах Saccharomyces cerivisae включают pYepSec1 (Baldari, et al., 1987. EMBO J. 6: 229-234), pMFa (Kuijan and Herskowitz, 1982. Cell 30: 933-943), pJRY88 (Schultz et al., 1987. Gene 54: 113-123), pYES2 (Invitrogen Corporation, Сан-Диего, Калифорния) и picZ (InVitrogen Corp, Сан-Диего, Калифорния).
В некоторых вариантах осуществления вектор управляет экспрессией белка в клетках насекомых при помощи бакуловирусных векторов экспрессии. Бакуловирусные векторы, доступные для экспрессии белков в культивируемых клетках насекомых (к примеру, клетках SF9), включают группу pAc (Smith, et al., 1983. Mol. Cell. Biol. 3: 2156-2165) и группу pVL (Lucklow and Summers, 1989. Virology 170: 31-39).
В некоторых вариантах осуществления вектор способен управлять экспрессией одной или нескольких последовательностей в клетках млекопитающих при помощи вектора экспрессии для млекопитающих. Примеры векторов экспрессии для млекопитающих включают pCDM8 (Seed, 1987. Nature 329: 840) и pMT2PC (Kaufman, et al., 1987. EMBO J. 6: 187-195). При использовании клеток млекопитающих функции контроля вектора экспрессии, как правило, обеспечиваются одним или несколькими регуляторными элементами. Например, широко используемые промоторы получают из вируса полиомы, аденовируса 2, цитомегаловируса, вируса обезьян 40 и других, раскрытых в данном документе и известных в уровне техники. Что качается других подходящих систем экспрессии как для прокариотических, так и для эукариотических клеток, см., к примеру, главы 16 и 17 в Sambrook, et al., MOLECULAR CLONING: A LABORATORY MANUAL. 2nd ed., Cold Spring Harbor Laboratory, Cold Spring Harbor Laboratory Press, Cold Spring Harbor, N.Y., 1989.
В некоторых вариантах осуществления рекомбинантные векторы экспрессии для млекопитающих способны управлять экспрессией нуклеиновой кислоты преимущественно в определенном типе клеток (к примеру, тканеспецифичные регуляторные элементы используют для экспрессии нуклеиновой кислоты). Тканеспецифичные регуляторные элементы известны из уровня техники. Неограничивающие примеры подходящих тканеспецифичных промоторов включают промотор гена альбумина (специфичный к печени; Pinkert, et al., 1987. Genes Dev. 1: 268-277), специфичные к лимфоидной ткани промоторы (Calame and Eaton, 1988. Adv. Immunol. 43: 235-275), в частности, промоторы рецепторов T-клеток (Winoto and Baltimore, 1989. EMBO J. 8: 729-733) и иммуноглобулины (Baneiji, et al., 1983. Cell 33: 729-740; Queen and Baltimore, 1983. Cell 33: 741-748), нейрон-специфичные промоторы (к примеру, промотор гена нейрофиламента; Byrne and Ruddle, 1989. Proc. Natl. Acad. Sci. USA 86: 5473-5477), специфичные к клеткам поджелудочной железы промоторы (Edlund, et al., 1985. Science 230: 912-916) и специфичные к клеткам молочной железы промоторы (к примеру, промотор молочной сыворотки; патент США № 4873316 и публикация европейской заявки № 264166). Регулируемые стадией развития промоторы также охвачены, к примеру, промоторы генов hox мыши (Kessel and Gruss, 1990. Science 249: 374-379) и промотор гена α-фетопротеина (Campes and Tilghman, 1989. Genes Dev. 3: 537-546).
В некоторых вариантах осуществления регуляторный элемент является функционально связанным с одним или несколькими элементами системы CRISPR так, чтобы управлять экспрессией одного или нескольких элементов системы CRISPR. В целом, CRISPR (короткие палиндромные повторы, регулярно расположенные группами), также известные как SPIDR (чередующиеся со спейсерами прямые повторы), составляют семейство локусов ДНК, которые, как правило, специфичны для определенного вида бактерий. Локус CRISPR включает определенный класс чередующихся коротких повторов последовательностей (SSR), которые были обнаружены у E. coli (Ishino et al., J. Bacteriol., 169:5429-5433 [1987]; и Nakata et al., J. Bacteriol., 171:3553-3556 [1989]), и ассоциированные гены. Подобные чередующиеся SSR были идентифицированы у Haloferax mediterranei, Streptococcus pyogenes, Anabaena и Mycobacterium tuberculosis (см., Groenen et al., Mol. Microbiol., 10:1057-1065 [1993]; Hoe et al., Emerg. Infect. Dis., 5:254-263 [1999]; Masepohl et al., Biochim. Biophys. Acta 1307:26-30 [1996]; и Mojica et al., Mol. Microbiol., 17:85-93 [1995]). Локусы CRISPR, как правило, отличаются от других SSR по структуре повторов, которые были названы короткими повторами с регулярными интервалами (SRSR) (Janssen et al., OMICS J. Integ. Biol., 6:23-33 [2002]; и Mojica et al., Mol. Microbiol., 36:244-246 [2000]). В целом, повторы являются короткими элементами, которые встречаются группами, которые регулярно разделены уникальными вставочными последовательностями с практически постоянной длинной (Mojica et al., [2000], выше). Несмотря на то, что последовательности повторов высоко консервативны между штаммами, некоторое количество чередующихся повторов и последовательностей спейсерных участков, как правило, отличаются от штамма к штамму (van Embden et al., J. Bacteriol., 182:2393-2401 [2000]). Локусы CRISPR были идентифицированы у более чем 40 видов прокариот (см., к примеру, Jansen et al., Mol. Microbiol., 43:1565-1575 [2002]; и Mojica et al., [2005]), в том числе, без ограничения, Aeropyrum, Pyrobaculum, Sulfolobus, Archaeoglobus, Halocarcula, Methanobacterium, Methanococcus, Methanosarcina, Methanopyrus, Pyrococcus, Picrophilus, Thermoplasma, Corynebacterium, Mycobacterium, Streptomyces, Aquifex, Porphyromonas, Chlorobium, Thermus, Bacillus, Listeria, Staphylococcus, Clostridium, Thermoanaerobacter, Mycoplasma, Fusobacterium, Azarcus, Chromobacterium, Neisseria, Nitrosomonas, Desulfovibrio, Geobacter, Myxococcus, Campylobacter, Wolinella, Acinetobacter, Erwinia, Escherichia, Legionella, Methylococcus, Pasteurella, Photobacterium, Salmonella, Xanthomonas, Yersinia, Treponema и Thermotoga.
В целом, “система CRISPR” означает в совокупности транскрипты и другие элементы, участвующие в экспрессии CRISPR-ассоциированных (“Cas”) генов или управлении их активностью, в том числе последовательности, кодирующие ген Cas, tracr-(транс-активируемую CRISPR) последовательность (к примеру, tracrRNA или активную частичную tracrRNA), парную tracr-последовательность (охватывающую “прямой повтор” и tracrRNA-процессированный неполный прямой повтор в контексте эндогенной системы CRISPR), направляющую последовательность (также называемую “спейсером” в контексте эндогенной системы CRISPR) или другие последовательности и транскрипты с локуса CRISPR. В некоторых вариантах осуществления один или несколько элементов системы CRISPR получены из системы CRISPR I типа, II типа или III типа. В некоторых вариантах осуществления один или несколько элементов системы CRISPR получены из определенного организма, содержащего эндогенную систему CRISPR, как, например, Streptococcus pyogenes. В целом, система CRISPR характеризуется элементами, которые способствуют образованию комплекса CRISPR в сайте целевой последовательности (также называемой протоспейсером в контексте эндогенной системы CRISPR). В контексте образования комплекса CRISPR “целевая последовательность” означает последовательность, по отношению к которой направляющая последовательность разработана так, чтобы обладать комплементарностью, где гибридизация между целевой последовательностью и направляющей последовательностью способствует образованию комплекса CRISPR. Полная комплементарность не обязательна при условии, что имеет место достаточная комплементарность для осуществления гибридизации и способствования образованию комплекса CRISPR. Целевая последовательность может содержать любой полинуклеотид, как, например, ДНК- или РНК-полинуклеотиды. В некоторых вариантах осуществления целевая последовательность расположена в ядре или цитоплазме клетки. В некоторых вариантах осуществления целевая последовательность может находиться в органелле эукариотической клетки, например, митохондрии или хлоропласте. Последовательность или матрицу, которую можно применять для рекомбинации в целевом локусе, содержащем целевые последовательности, называют “матрицей редактирования”, или “полинуклеотидом для редактирования”, или “последовательностью для редактирования”. В аспектах настоящего изобретения экзогенный матричный полинуклеотид можно называть матрицей редактирования. В одном аспекте настоящего изобретения рекомбинация является гомологичной рекомбинацией.
Как правило, в контексте эндогенной системы CRISPR образование комплекса CRISPR (содержащего направляющую последовательность, гибридизирующуюся с целевой последовательностью и образующую комплекс с одним или несколькими белками Cas) приводит к расщеплению одной или обеих нитей в или около (к примеру, в пределах 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 20, 50 или более пар оснований от) целевой последовательности. Не вдаваясь в теорию полагают, что tracr-последовательность, которая может содержать или состоять из всей или части tracr-последовательности дикого типа (к примеру, приблизительно или более чем приблизительно 20, 26, 32, 45, 48, 54, 63, 67, 85 или более нуклеотидов tracr-последовательности дикого типа), может также образовывать часть комплекса CRISPR, как, например, путем гибридизации вдоль по меньшей мере части tracr-последовательности со всей или частью парной tracr-последовательности, которая функционально связана с направляющей последовательностью. В некоторых вариантах осуществления tracr-последовательность обладает достаточной комплементарностью с парной tracr-последовательностью для гибридизации и участия в образовании комплекса CRISPR. Как и в случае с целевой последовательностью, полагают, что полная комплементарность не является необходимой при условии, что она является достаточной для выполнения функции. В некоторых вариантах осуществления tracr-последовательность характеризуется по меньшей мере 50%, 60%, 70%, 80%, 90%, 95% или 99% комплементарности последовательности по длине парной tracr-последовательности при оптимальном выравнивании. В некоторых вариантах осуществления один или несколько векторов, управляющих экспрессией одного или нескольких элементов системы CRISPR, вводят в клетку-хозяина, так что экспрессия элементов системы CRISPR управляет образованием комплекса CRISPR на одном или нескольких целевых сайтах. Например, каждое из фермента Cas, направляющей последовательности, связанной с парной tracr-последовательностью, и tracr-последовательности может быть функционально связано с отдельными регуляторными элементами в отдельных векторах. В качестве альтернативы, два или более элементов, экспрессируемых с одних и тех же или различных регуляторных элементов, можно объединять в одном векторе, с одним или несколькими дополнительными векторами, обеспечивающими любые компоненты системы CRISPR, не включенные в первый вектор. Элементы системы CRISPR, которые объединены в один вектор, могут быть расположены в любой удобной ориентации, как, например, один элемент, расположенный 5’ (“выше”) относительно или 3’ (“ниже”) относительно второго элемента. Кодирующая последовательность одного элемента может быть расположена на той же или противоположной нити по отношению к кодирующей последовательности второго элемента и направлена в том же или противоположном направлении. В некоторых вариантах осуществления один промотор управляет экспрессией транскрипта, кодирующего фермент CRISPR, и одной или нескольких из направляющей последовательности, парной tracr-последовательности (необязательно функционально связанной с направляющей последовательностью) и tracr-последовательности, встроенных в одну или несколько интронных последовательностей (к примеру, каждая в разном интроне, две или более по меньшей мере в одном интроне или все в одном интроне). В некоторых вариантах осуществления фермент CRISPR, направляющая последовательность, парная tracr-последовательность и tracr-последовательность функционально связаны с одним и тем же промотором и экспрессируются с него. Одиночные векторные конструкции для SpCas9 показаны на фигуре 22.
В некоторых вариантах осуществления вектор содержит один или несколько сайтов встраивания, как, например, последовательность узнавания рестрикционной эндонуклеазой (также называемая “сайтом клонирования”). В некоторых вариантах осуществления один или несколько сайтов встраивания (к примеру, приблизительно или более чем приблизительно 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более сайтов встраивания) расположены выше и/или ниже одного или нескольких элементов последовательности одного или нескольких векторов. В некоторых вариантах осуществления вектор содержит сайт встраивания выше парной tracr-последовательности и необязательно ниже регуляторного элемента, функционально связанного с парной tracr-последовательностью, так что после встраивания направляющей последовательности в сайт встраивания и при экспрессии направляющая последовательность управляет специфичным к последовательности связыванием комплекса CRISPR с целевой последовательностью в эукариотической клетке. В некоторых вариантах осуществления вектор содержит два или более сайта встраивания, при этом каждый сайт встраивания расположен между двумя парными tracr-последовательностями с тем, чтобы обеспечить возможность встраивания направляющей последовательности в каждый сайт. При таком расположении две или более направляющих последовательностей могут содержать две или более копий одной направляющей последовательности, две или более различных направляющих последовательностей или их комбинации. В тех случаях, когда применяют несколько различных направляющих последовательностей, можно использовать одну экспрессирующую конструкцию для целенаправленного воздействия активности CRISPR на несколько различных соответствующих целевых последовательностей в клетке. Например, один вектор может содержать приблизительно или более чем приблизительно 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 15, 20 или более направляющих последовательностей. В некоторых вариантах осуществления приблизительно или более чем приблизительно 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более таких содержащих направляющие последовательности векторов могут быть предусмотрены и необязательно доставлены в клетку.
В некоторых вариантах осуществления вектор содержит регуляторный элемент, функционально связанный с кодирующей фермент последовательностью, кодирующей фермент CRISPR, как, например, белок Cas. Неограничивающие примеры белков Cas включают Cas1, Cas1B, Cas2, Cas3, Cas4, Cas5, Cas6, Cas7, Cas8, Cas9 (также известный как Csn1 и Csx12), Cas10, Csy1, Csy2, Csy3, Cse1, Cse2, Csc1, Csc2, Csa5, Csn2, Csm2, Csm3, Csm4, Csm5, Csm6, Cmr1, Cmr3, Cmr4, Cmr5, Cmr6, Csb1, Csb2, Csb3, Csx17, Csx14, Csx10, Csx16, CsaX, Csx3, Csx1, Csx15, Csf1, Csf2, Csf3, Csf4, их гомологи или их модифицированные варианты. Эти ферменты известны; например, аминокислотную последовательность белка Cas9 S. pyogenes можно найти в базе данных SwissProt под номером доступа Q99ZW2. В некоторых вариантах осуществления немодифицированный фермент CRISPR характеризуется активностью для расщепления ДНК, как, например, Cas9. В некоторых вариантах осуществления фермент CRISPR представляет собой Cas9, и им может быть Cas9 из S. pyogenes или S. pneumoniae. В некоторых вариантах осуществления фермент CRISPR управляет расщеплением одной или обеих нитей в определенной точке целевой последовательности, как, например, в пределах целевой последовательности и/или в пределах комплементарной последовательности целевой последовательности. В некоторых вариантах осуществления фермент CRISPR управляет расщеплением одной или обеих нитей в пределах приблизительно 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 15, 20, 25, 50, 100, 200, 500 или более пар оснований от первого или последнего нуклеотида целевой последовательности. В некоторых вариантах осуществления вектор кодирует фермент CRISPR, который является мутированным по отношению к соответствующему ферменту дикого типа, так что у мутированного фермента CRISPR отсутствует способность расщеплять одну или обе нити целевого полинуклеотида, содержащего целевую последовательность. Например, замена аспартата на аланин (D10A) в каталитическом домене RuvC I Cas9 из S. pyogenes трансформирует Cas9 из нуклеазы, которая расщепляет обе нити, в никазу (расщепляет одну нить). Другие примеры мутаций, которые превращают Cas9 в никазу, включают, без ограничения, H840A, N854A и N863A. В аспектах настоящего изобретения никазы можно применять для редактирования генома посредством гомологичной рекомбинации, например, на фигуре 21 показано редактирование генома посредством гомологичной рекомбинации. На фигуре 21 (a) показано схематическое изображение никазы SpCas9 с мутацией D10A в каталитическом домене RuvC I. (b) Схематическое представление гомологичной рекомбинации (HR) в локусе EMX1 человека при использовании смысловых или антисмысловых однонитевых олигонуклеотидов в качестве матриц для репарации. (c) Последовательность участка, модифицированного с помощью HR. (d) Анализ вставок/делеций в целевом локусе 1 EMX1 (n=3), опосредованных SpCas9 дикого типа (wt) и никазой SpCas9 (D10A), с помощью SURVEYOR. Стрелки указывают положения фрагментов ожидаемого размера.
В некоторых вариантах осуществления никазу Cas9 можно использовать в комбинации с направляющей(ими) последовательностью(ями), к примеру, двумя направляющими последовательностями, которые целенаправленно воздействуют, соответственно, на смысловую и антисмысловую нити ДНК-мишени. Эта комбинация позволяет надрезать обе нити и использовать их для индукции NHEJ. Авторы данной заявки показали (данные не показаны) эффективность двух мишеней для никаз (т. е. sgRNA, нацеленных на одну и ту же точку, но на различные нити ДНК) при индуцировании мутагенного NHEJ. Одиночная никаза (Cas9-D10A с одной sgRNA) не способна индуцировать NHEJ и создавать вставки/делеции, но авторы данной заявки показали, что двойная никаза (Cas9-D10A и две sgRNA, нацеленные на различные нити в одной и той же точке) способна делать это в эмбриональных стволовых клетках человека (hESC). Эффективность составляет приблизительно 50% таковой нуклеазы (т. е. нормального Cas9 без мутации D10) в hESC.
В качестве дополнительного примера два или более каталитических доменов Cas9 (RuvC I, RuvC II и RuvC III) можно подвергать мутациям с получением мутированного Cas9, у которого практически отсутствует вся активность для расщепления ДНК. В некоторых вариантах осуществления мутацию D10A объединяют с одной или несколькими из мутаций H840A, N854A или N863A с получением фермента Cas9, у которого практически отсутствует вся активность для расщепления ДНК. В некоторых вариантах осуществления фермент CRISPR рассматривают как такой, у которого практически отсутствует вся активность для расщепления ДНК, в случаях, когда активность для расщепления ДНК мутированного фермента составляет менее приблизительно 25%, 10%, 5%, 1%, 0,1%, 0,01% или меньше по отношению к его не мутированной форме. Могут быть целесообразными другие мутации; в тех случаях, когда Cas9 или другой фермент CRISPR получен из вида, отличного от S. pyogenes, могут быть произведены мутации в соответствующих аминокислотах для достижения подобных эффектов.
В некоторых вариантах осуществления кодирующая фермент последовательность, кодирующая фермент CRISPR, является кодон-оптимизированной для экспрессии в определенных клетках, как, например, эукариотических клетках. Эукариотические клетки могут быть клетками определенного организма или полученными из него, как, например, млекопитающего, в том числе, без ограничения, человека, мыши, крысы, кролика, собаки или отличного от человека примата. В целом, оптимизация кодонов означает способ модификации последовательности нуклеиновой кислоты для повышения экспрессии в представляющих интерес клетках-хозяевах путем замещения по меньшей мере одного кодона (к примеру, приблизительно или более чем приблизительно 1, 2, 3, 4, 5, 10, 15, 20, 25, 50 или более кодонов) нативной последовательности кодонами, которые чаще или наиболее часто используют в генах этой клетки-хозяина, в то же время сохраняя нативную аминокислотную последовательность. Разные виды проявляют определенное "предпочтение" в отношении конкретных кодонов определенной аминокислоты. "Предпочтение" кодонов (различия в частоте использования кодонов между организмами) часто соотносят с эффективностью трансляции матричной РНК (мРНК), которая, в свою очередь, как полагают, зависит, среди прочего, от свойств кодонов, которые транслируются, и доступности конкретных молекул транспортной РНК (тРНК). Преобладание выбранных тРНК в клетке, как правило, является отражением кодонов, используемых наиболее часто при синтезе пептидов. Соответственно, гены могут быть приспособлены для оптимальной экспрессии генов в данном организме с использованием оптимизации кодонов. Таблицы частоты использования кодонов общедоступны, например, в "Базе данных частот использования кодонов" (“Codon Usage Database”), и эти таблицы можно адаптировать различными способами. См. Nakamura, Y., et al. “Codon usage tabulated from the international DNA sequence databases: status for the year 2000” Nucl. Acids Res. 28:292 (2000). Также доступны компьютерные алгоритмы для оптимизации кодонов определенной последовательности для экспрессии в определенной клетке-хозяине, как, например, также доступный Gene Forge (Aptagen; Джакобус, Пенсильвания). В некоторых вариантах осуществления один или несколько кодонов (к примеру, 1, 2, 3, 4, 5, 10, 15, 20, 25, 50 или более или все кодоны) в последовательности, кодирующей фермент CRISPR, соответствуют наиболее часто используемому кодону для определенной аминокислоты.
В целом, направляющая последовательность представляет собой любую полинуклеотидную последовательность, обладающую достаточной комплементарностью с целевой полинуклеотидной последовательностью для гибридизации с целевой последовательностью и управления специфичным к последовательности связыванием комплекса CRISPR с целевой последовательностью. В некоторых вариантах осуществления степень комплементарности между направляющей последовательностью и ее соответствующей целевой последовательностью при оптимальном выравнивании с использованием подходящего алгоритма выравнивания составляет приблизительно или более чем приблизительно 50%, 60%, 75%, 80%, 85%, 90%, 95%, 97,5%, 99% или более. Оптимальное выравнивание можно определять при помощи любого подходящего алгоритма для выравниваемых последовательностей, неограничивающие примеры которого включают алгоритм Смита-Ватермана, алгоритм Нидлмана-Вунша, алгоритмы, основанные на преобразовании Барроуза-Уилера (к примеру, Burrows Wheeler Aligner), ClustalW, Clustal X, BLAT, Novoalign (Novocraft Technologies), ELAND (Illumina, Сан-Диего, Калифорния), SOAP (доступный на soap.genomics.org.cn) и Maq (доступный на maq.sourceforge.net). В некоторых вариантах осуществления направляющая последовательность составляет приблизительно или более чем приблизительно 5, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 75 или более нуклеотидов в длину. В некоторых вариантах осуществления направляющая последовательность составляет менее чем приблизительно 75, 50, 45, 40, 35, 30, 25, 20, 15, 12 или менее нуклеотидов в длину. Способность направляющей последовательности управлять специфичным к последовательности связыванием комплекса CRISPR с целевой последовательностью можно оценить при помощи любого подходящего анализа. Например, компоненты системы CRISPR, достаточные для образования комплекса CRISPR, в том числе направляющая последовательность, которую необходимо исследовать, могут быть доставлены в клетку-хозяина с соответствующей целевой последовательностью, как, например, при помощи трансфекции векторами, кодирующими компоненты последовательности CRISPR, с последующей оценкой предпочтительного расщепления в пределах целевой последовательности, как, например, при помощи анализа с помощью Surveyor, который описан в данном документе. Подобным образом, расщепление целевой полинуклеотидной последовательности может быть определено в пробирке путем обеспечения целевой последовательности, компонентов комплекса CRISPR, в том числе направляющей последовательности, которую необходимо исследовать, и контрольной направляющей последовательности, отличной от тестовой направляющей последовательности, и сравнения воздействий тестовой и контрольной направляющей последовательности на связывание или скорость расщепления целевой последовательности. Другие анализы возможны и будут очевидны специалисту в данной области техники.
Направляющая последовательность может быть выбрана для целенаправленного воздействия на любую целевую последовательность. В некоторых вариантах осуществления целевая последовательность является последовательностью в пределах генома клетки. Иллюстративные целевые последовательности включают те, которые являются уникальными в целевом геноме. Например, для Cas9 S. pyogenes уникальная целевая последовательность в геноме может включать целевой сайт для Cas9 в виде MMMMMMMMNNNNNNNNNNNNXGG, где NNNNNNNNNNNNXGG (N представляет собой A, G, T или C; и X может быть любым) характеризуется единичным появлением в геноме. Уникальная целевая последовательность в геноме может включать целевой сайт для Cas9 S. pyogenes в виде MMMMMMMMMNNNNNNNNNNNXGG, где NNNNNNNNNNNXGG (N представляет собой A, G, T или C; и X может быть любым) характеризуется единичным появлением в геноме. Для Cas9 CRISPR1 S. thermophilus уникальная целевая последовательность в геноме может включать целевой сайт для Cas9 в виде MMMMMMMMNNNNNNNNNNNNXXAGAAW (SEQ ID NO: 1), где NNNNNNNNNNNNXXAGAAW (SEQ ID NO: 2) (N представляет собой A, G, T или C; X может быть любым; а W представляет собой A или T) характеризуется единичным появлением в геноме. Уникальная целевая последовательность в геноме может включать целевой сайт для Cas9 CRISPR1 S. thermophilus в виде MMMMMMMMMNNNNNNNNNNNXXAGAAW (SEQ ID NO: 3), где NNNNNNNNNNNXXAGAAW (SEQ ID NO: 4) (N представляет собой A, G, T или C; X может быть любым; а W представляет собой A или T) характеризуется единичным появлением в геноме. Для Cas9 S. pyogenes уникальная целевая последовательность в геноме может включать целевой сайт для Cas9 в виде MMMMMMMMNNNNNNNNNNNNXGGXG, где NNNNNNNNNNNNXGGXG (N представляет собой A, G, T или C; и X может быть любым) характеризуется единичным появлением в геноме. Уникальная целевая последовательность в геноме может включать целевой сайт для Cas9 S. pyogenes в виде MMMMMMMMMNNNNNNNNNNNXGGXG, где NNNNNNNNNNNXGGXG (N представляет собой A, G, T или C; и X может быть любым) характеризуется единичным появлением в геноме. В каждой из этих последовательностей “M” может представлять собой A, G, T или C и не должен учитываться при идентификации последовательности как уникальной.
В некоторых вариантах осуществления направляющая последовательность выбрана для снижения доли вторичной структуры в направляющей последовательности. Вторичную структуру можно определить при помощи любого подходящего алгоритма сворачивания полинуклеотида. Некоторые программы основаны на вычислении минимальной свободной энергии Гиббса. Примером одного такого алгоритма является mFold, который описан Zuker и Stiegler (Nucleic Acids Res. 9 (1981), 133-148). Другим примером алгоритма сворачивания является доступный в режиме онлайн веб-сервер RNAfold, разработанный в Институте теоретической химии при Венском университете, в котором используется алгоритм прогнозирования структуры на основе центроидного метода (см., к примеру, A.R. Gruber et al., 2008, Cell 106(1): 23-24; и PA Carr and GM Church, 2009, Nature Biotechnology 27(12): 1151-62). Дополнительные алгоритмы можно найти в заявке на патент США с серийным номером 61/836080 (номер дела у патентного поверенного 44790.11.2022; общая ссылка BI-2013/004A); включенной в данный документ при помощи ссылки.
В общем, парная tracr-последовательность включает любую последовательность, которая характеризуется достаточной комплементарностью с tracr-последовательностью для содействия одному или нескольким из следующих: (1) вырезания направляющей последовательности, фланкированной парными tracr-последовательностями, в клетке, содержащей соответствующую tracr-последовательность; и (2) образования комплекса CRISPR на целевой последовательности, где комплекс CRISPR содержит парную tracr-последовательность, гибридизирующуюся с tracr-последовательностью. В общем, степень комплементарности указана на основании оптимального выравнивания парной tracr-последовательности и tracr-последовательности по длине более короткой из двух последовательностей. Оптимальное выравнивание можно определить при помощи любого подходящего алгоритма выравнивания и можно дополнительно рассчитать для вторичных структур, как, например, самокомплементарность в пределах либо tracr-последовательности, либо парной tracr-последовательности. В некоторых вариантах осуществления степень комплементарности между tracr-последовательностью и парной tracr-последовательностью по длине более короткой из двух при оптимальном выравнивании составляет приблизительно или более чем приблизительно 25%, 30%, 40%, 50%, 60%, 70%, 80%, 90%, 95%, 97,5%, 99% или более. Примерные иллюстрации оптимального выравнивания между tracr-последовательностью и парной tracr-последовательностью представлены на фигурах 10B и 11B. В некоторых вариантах осуществления tracr-последовательность составляет приблизительно или более чем приблизительно 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 40, 50 или более нуклеотидов в длину. В некоторых вариантах осуществления tracr-последовательность и парная tracr-последовательность содержатся в одном транскрипте, так что гибридизация между ними двумя дает транскрипт со вторичной структурой, такой как "шпилька". Предпочтительные петлеобразующие последовательности для использования в "шпилечных" структурах составляют четыре нуклеотида в длину и наиболее предпочтительно имеют последовательность GAAA. Однако, можно использовать более короткие или длинные последовательности петли, а также альтернативные последовательности. Последовательности предпочтительно включают нуклеотидный триплет (например, AAA) и дополнительный нуклеотид (например, C или G). Примеры петлеобразующих последовательностей включают CAAA и AAAG. В одном варианте осуществления настоящего изобретения транскрипт или транскрибированная полинуклеотидная последовательность характеризуются по меньшей мере двумя или более "шпильками". В предпочтительных вариантах осуществления транскрипт характеризуется двумя, тремя, четырьмя или пятью "шпильками". В дополнительном варианте осуществления настоящего изобретения транскрипт характеризуется самое большее пятью "шпильками". В некоторых вариантах осуществления один транскрипт дополнительно включает последовательность терминации транскрипции; предпочтительно она является полиТ-последовательностью, например, из шести нуклеотидов T. Примерная иллюстрация такой "шпилечной" структуры представлена в нижней части фигуры 11B, где часть последовательности в 5’ направлении по отношению к концевому “N” и выше петли соответствует парной tracr-последовательности, а часть последовательности в 3’ направлении по отношению к петле соответствует tracr-последовательности. Дополнительными неограничивающими примерами отдельных полинуклеотидов, содержащих направляющую последовательность, парную tracr-последовательность и tracr-последовательность, являются следующие (перечисленные от 5’ к 3’), где “N” представляет собой основание направляющей последовательности, первый блок букв нижнего регистра представляет собой парную tracr-последовательность, а второй блок букв нижнего регистра представляет собой tracr-последовательность, и конечная поли-T-последовательность представляет собой терминатор транскрипции: (1) NNNNNNNNNNNNNNNNNNNNgtttttgtactctcaagatttaGAAAtaaatcttgcagaagctacaaagataaggcttcatgccgaaatcaacaccctgtcattttatggcagggtgttttcgttatttaaTTTTTT (SEQ ID NO: 5); (2) NNNNNNNNNNNNNNNNNNNNgtttttgtactctcaGAAAtgcagaagctacaaagataaggcttcatgccgaaatcaacaccctgtcattttatggcagggtgttttcgttatttaaTTTTTT (SEQ ID NO: 6); (3) NNNNNNNNNNNNNNNNNNNNgtttttgtactctcaGAAAtgcagaagctacaaagataaggcttcatgccgaaatcaacaccctgtcattttatggcagggtgtTTTTTT (SEQ ID NO: 7); (4) NNNNNNNNNNNNNNNNNNNNgttttagagctaGAAAtagcaagttaaaataaggctagtccgttatcaacttgaaaaagtggcaccgagtcggtgcTTTTTT (SEQ ID NO: 8); (5) NNNNNNNNNNNNNNNNNNNNgttttagagctaGAAATAGcaagttaaaataaggctagtccgttatcaacttgaaaaagtgTTTTTTT (SEQ ID NO: 9) и (6) NNNNNNNNNNNNNNNNNNNNgttttagagctagAAATAGcaagttaaaataaggctagtccgttatcaTTTTTTTT (SEQ ID NO: 10). В некоторых вариантах осуществления последовательности (1)-(3) используют в комбинации с Cas9 из CRISPR1 S. thermophilus. В некоторых вариантах осуществления последовательности (4)-(6) используют в комбинации с Cas9 из S. pyogenes. В некоторых вариантах осуществления tracr-последовательность является транскриптом, отдельным от транскрипта, содержащего парную tracr-последовательность (как, например, показанная в верхней части фигуры 11B).
В некоторых вариантах осуществления фермент CRISPR является частью слитого белка, содержащего один или несколько доменов гетерологичного белка (к примеру, приблизительно или более чем приблизительно 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более доменов в дополнение к ферменту CRISPR). Слитый белок, содержащий фермент CRISPR, может содержать любую дополнительную последовательность белка и необязательно линкерную последовательность между любыми двумя доменами. Примеры белковых доменов, которые могут быть слиты с ферментом CRISPR, включают, без ограничения, эпитопные метки, последовательности генов-репортеров и белковые домены с одним или несколькими из следующих видов активности: метилазной активности, деметилазной активности, активности для активации транскрипции, активности для репрессии транскрипции, активности фактора освобождения при транскрипции, активности для модификации гистонов, активности для расщепления ДНК и активности для связывания нуклеиновой кислоты. Неограничивающие примеры эпитопных меток включают гистидиновые (His) метки, V5-метки, FLAG-метки, метки гемагглютинина вируса гриппа (HA), Myc-метки, VSV-G-метки и тиоредоксиновые (Trx) метки. Примеры генов-репортеров включают, без ограничения, глутатион-S-трансферазу (GST), пероксидазу хрена (HRP), хлорамфеникол-ацетилтрансферазу (CAT), бета-галактозидазу, бета-глюкуронидазу, люциферазу, зеленый флуоресцентный белок (GFP), HcRed, DsRed, голубой флуоресцентный белок (CFP), желтый флуоресцентный белок (YFP) и автофлуорисцирующие белки, в том числе синий флуоресцентный белок (BFP). Фермент CRISPR может быть слит с последовательностью гена, кодирующей белок или фрагмент белка, которые связываются с молекулой ДНК или связываются с другими клеточными молекулами, в том числе, без ограничения, связывающий мальтозу белок (MBP), S-метку, продукты слияния Lex A и ДНК-связывающего домена (DBD), продукты слияния GAL4 и ДНК-связывающего домена и продукты слияния белка BP16 вируса простого герпеса (HSV). Дополнительные домены, которые могут образовывать часть слитого белка, содержащего фермент CRISPR, описаны в US20110059502, включенном в данный документ при помощи ссылки. В некоторых вариантах осуществления меченый фермент CRISPR используют для идентификации расположения целевой последовательности.
В одном аспекте настоящего изобретения ген-репортер, который включает, без ограничения, глутатион-S-трансферазу (GST), пероксидазу хрена (HRP), хлорамфеникол-ацетилтрансферазу (CAT) бета-галактозидазу, бета-глюкуронидазу, люциферазу, зеленый флуоресцентный белок (GFP), HcRed, DsRed, голубой флуоресцентный белок (CFP), желтый флуоресцентный белок (YFP) и автофлуоресцирующие белки, в том числе синий флуоресцентный белок (BFP), может быть введен в клетку, чтобы кодировать продукт гена, который служит в качестве маркера, по которому измеряют изменение или модификацию экспрессии продукта гена. В дополнительном варианте осуществления настоящего изобретения молекула ДНК, кодирующая продукт гена, может быть введена в клетку с помощью вектора. В предпочтительном варианте осуществления настоящего изобретения продуктом гена является люцифераза. В дополнительном варианте осуществления настоящего изобретения экспрессия продукта гена снижается.
В некоторых аспектах настоящее изобретение предусматривает способы, включающие доставку одного или нескольких полинуклеотидов, как, например, или одного или нескольких векторов, которые описаны в данном документе, одного или нескольких их транскриптов и/или одного или нескольких белков, транскрибируемых с них, в клетку-хозяина. Настоящее изобретение служит базисом для обеспечения целенаправленной модификации геномов на основе ДНК. Оно может быть совместимо со многими системами доставки, включая, без ограничения, вирусные, липосому, электропорацию, микроинъекцию и конъюгацию. В некоторых аспектах настоящее изобретение дополнительно предусматривает клетки, полученные при помощи таких способов, и организмы (такие как животные, растения или грибы), содержащие такие клетки или полученные из них. В некоторых вариантах осуществления фермент CRISPR в комбинации с (и необязательно образующий комплекс с) направляющей последовательностью доставляют в клетку. Традиционные способы переноса генов с использованием вирусов и без использования вирусов можно применять для введения нуклеиновых кислот в клетки млекопитающих или целевые ткани. Такие способы можно использовать для введения нуклеиновых кислот, кодирующих компоненты системы CRISPR, в клетки в культуре и в организме-хозяине. Системы доставки на основе векторов, отличных от вирусных, включают ДНК-плазмиды, РНК (к примеру, транскрипт вектора, описанного в данном документе), "оголенную" нуклеиновую кислоту и нуклеиновую кислоту, образующую комплекс со средством доставки, например, липосому. Системы доставки на основе вирусного вектора включают ДНК- и РНК-вирусы, которые имеют либо эписомальные, либо интегрированные геномы после доставки в клетку. В отношении обзора процедур генной терапии см. Anderson, Science 256:808-813 (1992); Nabel & Felgner, TIBTECH 11:211-217 (1993); Mitani & Caskey, TIBTECH 11:162-166 (1993); Dillon, TIBTECH 11:167-175 (1993); Miller, Nature 357:455-460 (1992); Van Brunt, Biotechnology 6(10):1149-1154 (1988); Vigne, Restorative Neurology and Neuroscience 8:35-36 (1995); Kremer & Perricaudet, British Medical Bulletin 51(1):31-44 (1995); Haddada et al., в Current Topics in Microbiology and Immunology, Doerfler and Böhm (eds) (1995); и Yu et al., Gene Therapy 1:13-26 (1994).
Способы отличной от вирусной доставки нуклеиновых кислот включают липофекцию, нуклеофекцию, микроинъекцию, баллистическую трансфекцию, виросомы, липосомы, иммунолипосомы, поликатион или конъюгаты липид:нуклеиновая кислота, "оголенную" ДНК, искусственные вирионы и повышенное при помощи средства поглощение ДНК. Липофекция описана, например, в патентах США №№ 5049386, 4946787 и 4897355), и реагенты для липофекции реализуются в промышленных масштабах (к примеру, Transfectam™ и Lipofectin™). Катионные и нейтральные липиды, которые подходят для эффективной липофекции полинуклеотидов с узнаванием рецептора, включают таковые из Felgner, WO 91/17424; WO 91/16024. Доставка может осуществляться в клетки (к примеру, in vitro или ex vivo введение) или целевые ткани (к примеру, in vivo введение).
Получение комплексов липид:нуклеиновая кислота, в том числе целенаправленно воздействующих липосом, как, например, иммунолипидных комплексов, хорошо известно специалистам в данной области (см., к примеру, Crystal, Science 270:404-410 (1995); Blaese et al., Cancer Gene Ther. 2:291-297 (1995); Behr et al., Bioconjugate Chem. 5:382-389 (1994); Remy et al., Bioconjugate Chem. 5:647-654 (1994); Gao et al., Gene Therapy 2:710-722 (1995); Ahmad et al., Cancer Res. 52:4817-4820 (1992); патенты США №№ 4186183, 4217344, 4235871, 4261975, 4485054, 4501728, 4774085, 4837028 и 4946787).
При применении систем на основе РНК- и ДНК-вирусов для доставки нуклеиновых кислот используют тщательно разработанные способы обеспечения целенаправленного воздействия вируса на конкретные клетки в организме и перемещения полезных последовательностей вируса в ядро. Вирусные векторы можно вводить непосредственно пациентам (in vivo) или их можно использовать для обработки клеток in vitro, и модифицированные клетки можно необязательно вводить пациентам (ex vivo). Традиционные системы на основе вирусов могут включать ретровирусные, лентивирусные, аденовирусные векторы, векторы на основе аденоассоциированного вируса и вируса простого герпеса для переноса генов. Интеграция в геном хозяина возможна с применением способов переноса генов на основе ретровируса, лентивируса и аденоассоциированного вируса, что часто приводит к длительной экспрессии встроенного трансгена. Кроме того, высокие показатели эффективности трансдукции наблюдали у многих различных типов клеток и целевых тканей.
Тропизм ретровирусов может быть изменен путем включения чужеродных белков оболочки с расширением возможной целевой популяции целевых клеток. Лентивирусные векторы являются ретровирусными векторами, которые способны трансфицировать или инфицировать неделящиеся клетки и, как правило, дают высокие вирусные титры. Выбор системы переноса генов на основе ретровирусов, таким образом, будет зависеть от целевой ткани. Ретровирусные векторы состоят из действующих в цис-положении длинных концевых повторов с упаковывающей способностью до 6-10 п.о. чужеродной последовательности. Минимальных действующих в цис-положении LTR достаточно для репликации и упаковки векторов, которые затем используют для интеграции терапевтического гена в целевую клетку с получением постоянной экспрессии трансгена. Широко применяемые ретровирусные векторы включают такие, которые основаны на вирусе лейкоза мышей (MuLV), вирусе лейкоза гиббонов (GaLV), вирусе иммунодефицита обезьян (SIV), вирусе иммунодефицита человека (HIV) и их комбинациях (см., к примеру, Buchscher et al., J. Virol. 66:2731-2739 (1992); Johann et al., J. Virol. 66:1635-1640 (1992); Sommnerfelt et al., Virol. 176:58-59 (1990); Wilson et al., J. Virol. 63:2374-2378 (1989); Miller et al., J. Virol. 65:2220-2224 (1991); PCT/US94/05700). В применениях, в которых транзиентная экспрессия является предпочтительной, можно применять системы на основе аденовирусов. Векторы на основе аденовирусов способны проявлять очень высокую эффективность трансдукции во многих типах клеток и не требуют деления клеток. С применением таких векторов были получены высокие титры и уровни экспрессии. Такой вектор можно получать в больших количествах в относительно простой системе. Векторы на основе аденоассоциированного вируса (“AAV”) также можно использовать для трансдукции клеток целевыми нуклеиновыми кислотами, к примеру, при получении in vitro нуклеиновых кислот и пептидов, и для процедур генной терапии in vivo и ex vivo (см., к примеру, West et al., Virology 160:38-47 (1987); патент США № 4797368; WO 93/24641; Kotin, Human Gene Therapy 5:793-801 (1994); Muzyczka, J. Clin. Invest. 94:1351 (1994). Создание рекомбинантных AAV-векторов описано в ряде публикаций, в том числе в патенте США № 5173414; Tratschin et al., Mol. Cell. Biol. 5:3251-3260 (1985); Tratschin, et al., Mol. Cell. Biol. 4:2072-2081 (1984); Hermonat & Muzyczka, PNAS 81:6466-6470 (1984); и Samulski et al., J. Virol. 63:03822-3828 (1989).
Упаковывающие клетки, как правило, используют для получения вирусных частиц, которые способны инфицировать клетку-хозяина. Такие клетки включают клетки 293, которые упаковывают аденовирус, и клетки ψ2 или клетки PA317, которые упаковывают ретровирус. Вирусные векторы, используемые в генной терапии, как правило, создают путем получения линии клеток, которые упаковывают вектор на основе нуклеиновой кислоты в вирусную частицу. Векторы обычно содержат минимальные вирусные последовательности, необходимые для упаковки и последующей интеграции в хозяина, при этом другие вирусные последовательности замещены кассетой экспрессии для экспрессии полинуклеотида(ов). Отсутствующие вирусные функции, как правило, обеспечивают во вспомогательном объекте при помощи линии упаковывающих клеток. Например, AAV-векторы, применяемые в генной терапии, как правило, имеют только ITR-последовательности из генома AAV, которые необходимы для упаковки и интеграции в геном хозяина. Вирусная ДНК упакована в линию клеток, которая содержит вспомогательную плазмиду, кодирующую другие гены AAV, а именно rep и cap, но без ITR-последовательностей. Линия клеток также может быть инфицирована аденовирусом в качестве вируса-помощника. Вирус-помощник способствует репликации AAV-вектора и экспрессии генов AAV из вспомогательной плазмиды. Вспомогательная плазмида не упаковывается в значительных количествах в связи с отсутствием ITR-последовательностей. Контаминация аденовирусом может быть снижена, к примеру, при помощи тепловой обработки, к которой аденовирус более чувствителен, чем AAV. Дополнительные способы доставки нуклеиновых кислот в клетки известны специалистам в данной области. См., например, US20030087817, включенный в данный документ при помощи ссылки.
В некоторых вариантах осуществления клетка-хозяин транзиентно или не транзиентно трасфицирована одним или несколькими векторами, описанными в данном документе. В некоторых вариантах осуществления клетка трансфицирована так, как это в естественных условиях происходит у субъекта. В некоторых вариантах осуществления клетка, которую трансфицируют, взята от субъекта. В некоторых вариантах осуществления клетка получена из клеток, взятых от субъекта, как, например, линии клеток. Широкий спектр линий клеток для культуры тканей известен в уровне техники. Примеры линий клеток включают, без ограничения, C8161, CCRF-CEM, MOLT, mIMCD-3, NHDF, HeLa-S3, Huh1, Huh4, Huh7, HUVEC, HASMC, HEKn, HEKa, MiaPaCell, Panc1, PC-3, TF1, CTLL-2, C1R, Rat6, CV1, RPTE, A10, T24, J82, A375, ARH-77, Calu1, SW480, SW620, SKOV3, SK-UT, CaCo2, P388D1, SEM-K2, WEHI-231, HB56, TIB55, Jurkat, J45.01, LRMB, Bcl-1, BC-3, IC21, DLD2, Raw264.7, NRK, NRK-52E, MRC5, MEF, Hep G2, HeLa B, HeLa T4, COS, COS-1, COS-6, COS-M6A, эпителиальные клетки почки обезьяны BS-C-1, эмбриональные фибробласты мыши BALB/ 3T3, 3T3 Swiss, 3T3-L1, фетальные фибробласты человека 132-d5; фибробласты мыши 10.1, 293-T, 3T3, 721, 9L, A2780, A2780ADR, A2780cis, A172, A20, A253, A431, A-549, ALC, B16, B35, клетки BCP-1, BEAS-2B, bEnd.3, BHK-21, BR 293, BxPC3, C3H-10T1/2, C6/36, Cal-27, CHO, CHO-7, CHO-IR, CHO-K1, CHO-K2, CHO-T, CHO Dhfr -/-, COR-L23, COR-L23/CPR, COR-L23/5010, COR-L23/R23, COS-7, COV-434, CML T1, CMT, CT26, D17, DH82, DU145, DuCaP, EL4, EM2, EM3, EMT6/AR1, EMT6/AR10.0, FM3, H1299, H69, HB54, HB55, HCA2, HEK-293, HeLa, Hepa1c1c7, HL-60, HMEC, HT-29, Jurkat, клетки JY, клетки K562, Ku812, KCL22, KG1, KYO1, LNCap, Ma-Mel 1-48, MC-38, MCF-7, MCF-10A, MDA-MB-231, MDA-MB-468, MDA-MB-435, MDCK II, MDCK II, MOR/0.2R, MONO-MAC 6, MTD-1A, MyEnd, NCI-H69/CPR, NCI-H69/LX10, NCI-H69/LX20, NCI-H69/LX4, NIH-3T3, NALM-1, NW-145, линии клеток OPCN / OPCT, Peer, PNT-1A / PNT 2, RenCa, RIN-5F, RMA/RMAS, клетки Saos-2, Sf-9, SkBr3, T2, T-47D, T84, линию клеток THP1, U373, U87, U937, VCaP, клетки Vero, WM39, WT-49, X63, YAC-1, YAR и их трансгенные варианты. Линии клеток доступны из ряда источников, известных специалистам в данной области (см., к примеру, Американскую коллекцию типовых культур (ATCC) (Манассас, Вирджиния)). В некоторых вариантах осуществления клетку, трансфицированную одним или несколькими векторами, описанными в данном документе, используют для получения новой линии клеток, содержащей одну или несколько полученных из вектора последовательностей. В некоторых вариантах осуществления клетку, транзиентно трансфицированную компонентами системы CRISPR, которая описана в данном документе (как, например, путем транзиентной трансфекции одним или несколькими векторами или трансфекции РНК), и модифицированную при помощи активности комплекса CRISPR, используют для получения новой линии клеток, содержащей клетки, которые содержат модификацию, но у которых отсутствует любая другая экзогенная последовательность. В некоторых вариантах осуществления клетки, транзиентно или не транзиентно трансфицированные одним или несколькими векторами, описанными в данном документе, или линии клеток, полученные из таких клеток, использовали при оценивании одного или нескольких тестовых соединений.
Аспекты настоящего изобретения касаются поколения изогенных линий клеток млекопитающих для изучения генетических вариаций при заболевании. Дополнительный аспект настоящего изобретения относится к поколению генетически модифицированных животных моделей, либо трансгенных, либо полученных с помощью вирус-опосредованной доставки. Настоящее изобретение также охватывает модификацию генома микроорганизмов, клеток, растений, животных или синтетических организмов для получения продуктов, полезных с точки зрения биомедицины, сельского хозяйства и промышленности. В еще одном аспекте настоящее изобретение охватывает генную терапию. Настоящее изобретение можно использовать в качестве инструмента биологического исследования, для составления представления о геноме, например, для исследований нокаута генов. Настоящее изобретение относится ко многим другим способам и композициям, которые зависят от базовой возможности редактирования и "переписывания" ДНК, содержащейся в геномах, а также целенаправленной инактивации организмов, имеющих ДНК. Настоящее изобретение также можно применять в качестве терапевтического средства для целенаправленного воздействия на определенные штаммы, вызывающие разновидности бактериальной инфекции, вирусную инфекцию и т.д.
В некоторых вариантах осуществления один или несколько векторов, описанных в данном документе, используют для получения отличного от человека трансгенного животного или трансгенного растения. В некоторых вариантах осуществления трансгенным животным является млекопитающее, как, например, мышь, крыса или кролик. В определенных вариантах осуществления организмом или субъектом является растение. В определенных вариантах осуществления организмом, или субъектом, или растением является водоросль. Способы получения трансгенных растений и животных известны в уровне техники и, как правило, начинаются со способа трансфекции клетки, такого как описанный в данном документе. Также представлены трансгенные животные, как и трансгенные растения, в частности, сельскохозяйственные культуры и водоросли. Трансгенное животное или растение могут быть полезными в других путях применения, помимо обеспечения модели заболевания. Они могут включать производство пищи или кормопроизводство посредством биосинтеза, например, белков, углеводов, питательных веществ или витаминов на более высоких уровнях, чем наблюдалось бы в обычных условиях у дикого типа. В этом отношении предпочтительными являются трансгенные растения, в особенности зернобобовые и клубнеплоды, и животные, в особенности млекопитающие, такие как крупный рогатый скот (коровы, овцы, козы и свиньи), но также домашняя птица и съедобные насекомые.
Трансгенные водоросли или другие растения, такие как рапс, могут быть особенно применимыми в производстве растительных масел или таких видов биотоплива, как, например, спирты (особенно метанол и этанол). Они могут быть сконструированы для синтеза или сверхсинтеза масла или спиртов на высоких уровнях для применения в масложировой или биотопливной промышленности.
В одном аспекте настоящее изобретение предусматривает способы модификации целевого полинуклеотида в эукариотической клетке, что может происходить in vivo, ex vivo или in vitro. В некоторых вариантах осуществления способ включает забор клетки или популяции клеток от человека, или отличного от человека животного, или растения (в том числе микроскопических водорослей) и модификацию клетки или клеток. Культивирование можно осуществлять на любой стадии ex vivo. Клетку или клетки можно даже повторно вводить отличному от человека животному или в растение (в том числе микроскопические водоросли).
В одном аспекте настоящее изобретение предусматривает способы модификации целевого полинуклеотида в эукариотической клетке. В некоторых вариантах осуществления способ включает обеспечение связывания комплекса CRISPR с целевым полинуклеотидом для осуществления расщепления указанного целевого полинуклеотида с модификацией, таким образом, целевого полинуклеотида, где комплекс CRISPR содержит фермент CRISPR, образующий комплекс с направляющей последовательностью, гибридизирующейся с целевой последовательностью в указанном целевом полинуклеотиде, где указанная направляющая последовательность связана с парной tracr-последовательностью, которая, в свою очередь, гибридизируется с tracr-последовательностью.
В одном аспекте настоящее изобретение предусматривает способ модификации экспрессии полинуклеотида в эукариотической клетке. В некоторых вариантах осуществления способ включает обеспечение связывания комплекса CRISPR с полинуклеотидом так, что указанное связывание приводит к повышенной или пониженной экспрессии указанного полинуклеотида; где комплекс CRISPR содержит фермент CRISPR, образующий комплекс с направляющей последовательностью, гибридизирующейся с целевой последовательностью в указанном целевом полинуклеотиде, где указанная направляющая последовательность связана с парной tracr-последовательностью, которая, в свою очередь, гибридизируется с tracr-последовательностью.
С учетом недавних достижений в области геномики сельскохозяйственных культур возможность применения системы CRISPR-Cas для осуществления эффективных и экономичных редактирования генов и манипуляции с ними обеспечит возможность быстрого отбора и сравнения одиночных и мультиплексных генетических манипуляций для трансформирования таких геномов в отношении повышенного производства и улучшенных признаков. В связи с этим делается ссылка на патенты США и публикации патентов США: патент США № 6603061 - опосредованный агробактериями способ трансформации растений (Agrobacterium-Mediated Plant Transformation Method); патент США № 7868149 - последовательности генома растений и их применение (Plant Genome Sequences and Uses Thereof) и US 2009/0100536 - трансгенные растения с улучшенными агротехническими признаками (Transgenic Plants with Enhanced Agronomic Traits), все содержания и раскрытия каждого из которых включены в данный документ при помощи ссылки в полном объеме. При осуществлении на практике настоящего изобретения содержание и раскрытие Morrell et al. “Crop genomics:advances and applications” Nat Rev Genet. 2011 Dec 29;13(2):85-96 также включены в данный документ при помощи ссылки в полном объеме.
У растений патогены часто являются специфичными по отношению к хозяину. Например, Fusarium oxysporum f. sp. lycopersici вызывает фузариозный вилт томата, но поражает только томат, а F. oxysporum f. dianthii и Puccinia graminis f. sp. tritici поражают только пшеницу. Растения обладают присущими и индуцированными защитными реакциями, обеспечивающие устойчивость к большинству патогенов. Мутации и события рекомбинации в поколениях растений приводят к генетической изменчивости, которая обуславливает восприимчивость, тем более, что патогены размножаются с большей частотой, чем растения. У растений может наблюдаться устойчивость видов-нехозяев, например, хозяин и патоген являются несовместимыми. Также может наблюдаться горизонтальная устойчивость, например, частичная устойчивость ко всем расам патогена, обычно контролируемая многими генами, и вертикальная устойчивость, например, полная устойчивость к некоторым расам патогена, но не к другим расам, обычно контролируемая несколькими генами. На уровне взаимодействия генов растения и патогены эволюционируют совместно, и генетические изменения одного уравновешивают изменения другого. Соответственно, используя естественную изменчивость, селекционеры комбинируют наиболее полезные гены для урожайности, качества, однородности, выносливости, устойчивости. Источники генов устойчивости включают нативные или чужеродные сорта, старинные сорта, родственные дикорастущие растения и индуцированные мутации, например, при обработке растительного материала мутагенными средствами. Применяя настоящее изобретение, селекционеры растений получают новый инструмент для индукции мутаций. Соответственно, специалист в данной области может проанализировать геном источников генов устойчивости, а в отношении сортов, имеющих желаемые характеристики или признаки, использовать настоящее изобретение для индукции появления генов устойчивости с большей точностью, чем в случае применявшихся ранее мутагенных средств, и, следовательно, ускорять и улучшать программы селекции растений.
В одном аспекте настоящее изобретение предусматривает наборы, содержащие любой один или несколько из элементов, раскрытых в приведенных выше способах и композициях. В некоторых вариантах осуществления набор содержит векторную систему и инструкции по применению набора. В некоторых вариантах осуществления векторная система содержит (a) первый регуляторный элемент, функционально связанный с парной tracr-последовательностью и одним или несколькими сайтами встраивания для встраивания направляющей последовательности выше парной tracr-последовательности, где при экспрессии направляющая последовательность управляет специфичным к последовательности связыванием комплекса CRISPR с целевой последовательностью в эукариотической клетке, где комплекс CRISPR содержит фермент CRISPR, образующий комплекс с (1) направляющей последовательностью, которая гибридизируется с целевой последовательностью, и (2) парной tracr-последовательностью, которая гибридизируется с tracr-последовательностью; и/или (b) второй регуляторный элемент, функционально связанный с кодирующей фермент последовательностью, кодирующей указанный фермент CRISPR, содержащий последовательность ядерной локализации. Элементы могут быть предоставлены отдельно или в комбинациях и могут быть предоставлены в любом подходящем контейнере, как, например, ампуле, флаконе или пробирке. В некоторых вариантах осуществления набор включает инструкции на одном или нескольких языках, например, на более чем одном языке.
В некоторых вариантах осуществления набор содержит один или несколько реагентов для применения в способе, в котором используется один или несколько элементов, описанных в данном документе. Реагенты могут быть предоставлены в любом подходящем контейнере. Например, набор может предусматривать один или несколько реакционных буферов или буферов для хранения. Реагенты могут быть предоставлены в форме, которая применима в конкретном анализе, или в форме, которая предусматривает добавление одного или нескольких других компонентов перед применением (к примеру, в форме концентрата или лиофилизированной форме). Буфер может быть любым буфером, в том числе, без ограничения, буфером с карбонатом натрия, буфером с бикарбонатом натрия, боратным буфером, Tris-буфером, буфером MOPS, буфером HEPES и их комбинациями. В некоторых вариантах осуществления буфер является щелочным. В некоторых вариантах осуществления буфер имеет значение pH от приблизительно 7 до приблизительно 10. В некоторых вариантах осуществления набор содержит один или несколько олигонуклеотидов, соответствующих направляющей последовательности, для встраивания в вектор для того, чтобы имела место функциональная связь направляющей последовательности и регуляторного элемента. В некоторых вариантах осуществления набор содержит матричный полинуклеотид для гомологичной рекомбинации.
В одном аспекте настоящее изобретение предусматривает способы применения одного или нескольких элементов системы CRISPR. Комплекс CRISPR по настоящему изобретению обеспечивает эффективное средство модификации целевого полинуклеотида. Комплекс CRISPR по настоящему изобретению характеризуется большим разнообразием полезных свойств, включая модификацию (например, делецию, вставку, транслокацию, инактивацию, активацию) целевого полинуклеотида во множестве типов клеток. Комплекс CRISPR по настоящему изобретению как таковой имеет широкий спектр применений, к примеру, в генной терапии, скрининге лекарственных средств, диагностике и прогнозировании заболеваний. Иллюстративный комплекс CRISPR содержит фермент CRISPR, образующий комплекс с направляющей последовательностью, которая гибридизируется с целевой последовательностью в целевом полинуклеотиде. Направляющая последовательность связана с парной tracr-последовательностью, которая, в свою очередь, гибридизируется с tracr-последовательностью.
Целевым полинуклеотидом комплекса CRISPR может быть любой полинуклеотид, эндогенный или экзогенный по отношению к эукариотической клетке. Например, целевым полинуклеотидом может быть полинуклеотид, находящийся в ядре эукариотической клетки. Целевой полинуклеотид может быть последовательностью, кодирующей продукт гена (к примеру, белок), или некодирующей последовательностью (к примеру, регуляторным полинуклеотидом или избыточной ДНК). Не желая быть связанными теорией, полагают, что целевая последовательность должна быть ассоциирована с PAM (мотивом, смежным с протоспейсером); то есть короткой последовательностью, узнаваемой комплексом CRISPR. Определенные требования в отношении последовательности и длины PAM различаются в зависимости от применяемого фермента CRISPR, но PAM, как правило, является последовательностью из 2-5 пар оснований, смежной с протоспейсером (то есть целевой последовательностью). Примеры последовательностей PAM приведены в разделе "Примеры" ниже, и специалист в данной области сможет выявить дополнительные последовательности PAM для применения с данным ферментом CRISPR.
Целевой полинуклеотид комплекса CRISPR может включать некоторое количество ассоциированных с заболеваниями генов и полинуклеотидов, а также генов и полинуклеотидов, ассоциированных с биохимическими путями передачи сигнала, которые перечислены в предварительных заявках на патент США 61/736527 и 61/748427 с общей ссылкой BI-2011/008/WSGR, номер в реестре 44063-701.101, и BI-2011/008/WSGR, номер в реестре 44063-701.102, соответственно, обе озаглавленные "СИСТЕМЫ, СПОСОБЫ И КОМПОЗИЦИИ ДЛЯ МАНИПУЛЯЦИИ С ПОСЛЕДОВАТЕЛЬНОСТЯМИ" (SYSTEMS METHODS AND COMPOSITIONS FOR SEQUENCE MANIPULATION), поданные 12 декабря 2012 г. и 2 января 2013 г., соответственно, причем содержание всех из которых включено в данный документ при помощи ссылки в полном объеме.
Примеры целевых полинуклеотидов включают последовательность, ассоциированную с биохимическими путями передачи сигнала, к примеру, ген или полинуклеотид ассоциированный с биохимическими путями передачи сигнала. Примеры целевых полинуклеотидов включают ассоциированные с заболеваниями гены или полинуклеотиды. “Ассоциированный с заболеванием” ген или полинуклеотид означает любой ген или полинуклеотид, который обеспечивает продукты транскрипции или трансляции на отклоняющемся от нормы уровне или в отклоняющейся от нормы форме в клетках, полученных из пораженных заболеванием тканей, по сравнению с тканями или клетками контроля без заболевания. Это может быть ген, который начинает экспрессироваться при ненормально высоком уровне; это может быть ген, который начинает экспрессироваться при ненормально низком уровне, где измененная экспрессия коррелирует с появлением и/или развитием заболевания. Ассоциированный с заболеванием ген также означает ген, несущий мутацию(и) или генетическое изменение, который непосредственно ответственен или находится в неравновесном сцеплении с геном(ами), который(е) ответственен(ны) за этиологию заболевания. Транскрибируемые или транслируемые продукты могут быть известными или неизвестными и могут быть на нормальном уровне или на отклоняющемся от нормального уровне.
Примеры ассоциированных с заболеваниями генов и полинуклеотидов доступны от Института генетической медицины Маккьюсика-Натанса (McKusick-Nathans Institute of Genetic Medicine) при Университете Джонса Хопкинса (Johns Hopkins University) (Балтимор, Мэриленд) и Национального центра биотехнологической информации (National Center for Biotechnology Information) при Национальной библиотеке медицины (National Library of Medicine) (Бетесда, Мэриленд), доступных во всемирной сети Интернет.
Примеры ассоциированных с заболеваниями генов и полинуклеотидов перечислены в таблицах A и B. Конкретная информация в отношении заболеваний доступна от Института генетической медицины Маккьюсика-Натанса (McKusick-Nathans Institute of Genetic Medicine) при Университете Джонса Хопкинса (Johns Hopkins University) (Балтимор, Мэриленд) и Национального центра биотехнологической информации (National Center for Biotechnology Information), Национальной библиотеки медицины (National Library of Medicine) (Бетесда, Мэриленд), доступных во всемирной сети Интернет. Примеры ассоциированных с биохимическими путями передачи сигнала генов и полинуклеотидов перечислены в таблице C.
Мутации в этих генах и путях могут приводить к продуцированию несоответствующих белков или белков в несоответствующих количествах, которые воздействуют на функцию. Дополнительные примеры генов, заболеваний и белков, таким образом, включены при помощи ссылки из предварительных заявок на патент США 61/736527, поданной 12 декабря 2012 г., и 61/748427, поданной 2 февраля 2013 г. Такие гены, белки и пути могут быть целевым полинуклеотидом комплекса CRISPR.
Таблица A
Таблица B
нарушения, связанные
с кровеносной
системой и
свертыванием крови
клеточной дисрегуляцией заболевания и
нарушения и
онкологические заболевания и
нарушения
воспалением и
иммунной системой заболевания и
нарушения
связанные с
обменом белка
заболевания и
нарушения
нарушения
нейрональные заболевания и
нарушения
нарушения
замедленным/
быстрым развитием (3)
Таблица C
участием PI3K/AKT
глюкокортикоидного рецептора
участием эфринового рецептора
цитоскелет
Гентингтона
участием B-клеточного рецептора
лейкоцитов
участием интегрина
участием PTEN
участием p53
участием SAPK/JNK
участием PPAr/RXR
участием NF-KB
участием нейрегулина
гликонеогенез
Варианты осуществления настоящего изобретения также касаются способов и композиций, связанных с нокаутированием генов, амплифицированием генов и репарацией конкретных мутаций, ассоциированных с нестабильностью ДНК-повторов и неврологическими нарушениями (Robert D. Wells, Tetsuo Ashizawa, Genetic Instabilities and Neurological Diseases, Second Edition, Academic Press, Oct 13, 2011 – Medical). Как было обнаружено, определенные аспекты последовательностей тандемных повторов ответственны за более двадцати заболеваний человека (New insights into repeat instability: role of RNA•DNA hybrids. McIvor EI, Polak U, Napierala M. RNA Biol. 2010 Sep-Oct;7(5):551-8). Система CRISPR-Cas может быть приспособлена для корректировки таких дефектов геномной нестабильности.
Дополнительный аспект настоящего изобретения относится к использованию системы CRISPR-Cas для корректирования дефектов в генах EMP2A и EMP2B, которые, как было обнаружено, ассоциированы с болезнью Лафора. Болезнь Лафора представляет собой аутосомно-рецессивное состояние, которое характеризуется прогрессирующей миоклонус-эпилепсией, которая может начинаться с эпелиптических приступов в подростковом возрасте. Некоторые случаи заболевания могут быть вызваны мутациями в генах, которые уже были выявлены. Заболевание вызывает приступы, мышечные спазмы, затрудненную ходьбу, слабоумие и, в конечном итоге, смерть. В настоящее время не существует терапии, которая показала эффективность против развития заболевания. На другие генетические расстройства, ассоциированные с эпилепсией, также можно целенаправленно воздействовать при помощи системы CRISPR-Cas, и лежащие в основе генетические механизмы дополнительно описаны в Genetics of Epilepsy and Genetic Epilepsies, edited by Giuliano Avanzini, Jeffrey L. Noebels, Mariani Foundation Paediatric Neurology:20; 2009).
В еще одном аспекте настоящего изобретения систему CRISPR-Cas можно использовать для корректировки офтальмологических дефектов, которые являются результатом нескольких генетических мутаций, дополнительно описанных в Genetic Diseases of the Eye, Second Edition, edited by Elias I. Traboulsi, Oxford University Press, 2012.
Некоторые дополнительные аспекты настоящего изобретения касаются корректирования дефектов, ассоциированных с широким спектром наследственных заболеваний, которые дополнительно описаны на веб-сайте Национальных институтов здравоохранения (National Institutes of Health) в тематическом подразделе "Наследственные заболевания" ("Genetic Disorders") (веб-сайт по адресу health.nih.gov/topic/GeneticDisorders). Наследственные заболевания головного мозга могут включать, без ограничения, адренолейкодистрофию, агенезию мозолистого тела, синдром Айкарди, синдром Альперса, болезнь Альцгеймера, синдром Барта, болезнь Баттена, CADASIL, мозжечковую дегенерацию, болезнь Фабри, синдром Герстмана-Штраусслера-Шейнкера, болезнь Гентингтона и другие связанные с триплетным повтором нарушения, болезнь Лея, синдром Леша-Найхана, болезнь Менкеса, типы митохондриальной миопатии и кольпоцефалию по критериям NINDS. Такие заболевания дополнительно описаны на веб-сайте Национальных институтов здравоохранения (National Institutes of Health) в тематическом подразделе "Наследственные заболевания головного мозга" ("Genetic Brain Disorders").
В некоторых вариантах осуществления состоянием может быть неоплазия. В некоторых вариантах осуществления, где состоянием является неоплазия, гены, на которые целенаправленно воздействуют, являются любыми из перечисленных в таблице A (в данном случае PTEN и так далее). В некоторых вариантах осуществления состоянием может быть возрастная дегенерация желтого пятна. В некоторых вариантах осуществления состоянием может быть шизофреническое нарушение. В некоторых вариантах осуществления состоянием может быть связанное с тринуклеотидным повтором нарушение. В некоторых вариантах осуществления состоянием может быть синдром ломкой X-хромосомы. В некоторых вариантах осуществления состоянием может быть связанное с секретазой нарушение. В некоторых вариантах осуществления состоянием может быть связанное с прионами нарушение. В некоторых вариантах осуществления состоянием может быть ALS. В некоторых вариантах осуществления состоянием может быть привыкание к наркотическим средствам. В некоторых вариантах осуществления состоянием может быть аутизм. В некоторых вариантах осуществления состоянием может быть болезнь Альцгеймера. В некоторых вариантах осуществления состоянием может быть воспаление. В некоторых вариантах осуществления состоянием может быть болезнь Паркинсона.
Примеры белков, ассоциированных с болезнью Паркинсона, включают, без ограничения, α-синуклеин, DJ-1, LRRK2, PINK1, паркин, UCHL1, синфилин-1 и NURR1.
Примеры связанных с привыканием белков могут включать, например, ABAT.
Примеры связанных с воспалением белков могут включать, например, моноцитарный хемоаттрактантный белок-1 (monocyte chemoattractant protein-1) (MCP1), кодируемый геном Ccr2, C-C рецептор хемокина 5 типа (C-C chemokine receptor type 5) (CCR5), кодируемый геном Ccr5, IgG-рецептор IIB (IgG receptor IIB) (FCGR2b, также называемый CD32), кодируемый геном Fcgr2b, или белок Fc-эпсилон-R1g (Fc epsilon R1g) (FCER1g), кодируемый геном Fcer1g.
Примеры ассоциированных с заболеваниями сердечно-сосудистой системы белков могут включать, например, IL1B (интерлейкин 1, бета (interleukin 1, beta)), XDH (ксантиндегидрогеназу (xanthine dehydrogenase)), TP53 (опухолевый белок p53 (tumor protein p53)), PTGIS (простагландин-I2(простациклин)-синтазу (prostaglandin I2 (prostacyclin) synthase)), MB (миоглобин (myoglobin)), IL4 (интерлейкин 4 (interleukin 4)), ANGPT1 (ангиопоэтин 1 (angiopoietin 1)), ABCG8 (АТФ-связывающую кассету, подсемейство G (WHITE), представитель 8 (ATP-binding cassette, sub-family G (WHITE), member 8)) или CTSK (катепсин K (cathepsin K)).
Примеры ассоциированных с болезнью Альцгеймера белков могут включать, например, белок, представляющий собой рецептор липопротеинов очень низкой плотности (very low density lipoprotein receptor protein) (VLDLR), кодируемый геном VLDLR, убиквитин-подобный модификатор-активирующий фермент 1 (ubiquitin-like modifier activating enzyme 1) (UBA1), кодируемый геном UBA1, или белок, являющийся каталитической субъединицей NEDD8-активирующего фермента E1 (NEDD8-activating enzyme E1 catalytic subunit protein) (UBE1C), кодируемый геном UBA3.
Примеры белков, ассоциированных с расстройствами аутистического спектра, могут включать, например, белок 1, ассоциированный с периферическим бензодиазепиновым рецептором (benzodiazapine receptor (peripheral) associated protein 1) (BZRAP1), кодируемый геном BZRAP1, белок, представитель 2 семейства AF4/FMR2 (AF4/FMR2 family представитель 2 protein) (AFF2), кодируемый геном AFF2 (также называемый MFR2), белок-аутосомный гомолог 1, ассоциированный с умственной отсталостью, связанной с ломкой X-хромосомой (fragile X mental retardation autosomal homolog 1 protein) (FXR1), кодируемый геном FXR1, или белок-аутосомный гомолог 2, ассоциированный с умственной отсталостью, связанной с ломкой X-хромосомой (fragile X mental retardation autosomal homolog 2 protein) (FXR2), кодируемый геном FXR2.
Примеры белков, ассоциированных с дегенерацией желтого пятна, могут включать, например, АТФ-связывающую кассету, белок-представитель 4 подсемейства A (ABC1) (ATP-binding cassette, sub-family A (ABC1) member 4 protein) (ABCA4), кодируемый геном ABCR, белок-аполипротеин E (apolipoprotein E protein) (APOE), кодируемый геном APOE, или белок-лиганд 2 хемокина (C-C мотив) (chemokine (C-C motif) Ligand 2 protein) (CCL2), кодируемый геном CCL2.
Примеры белков, ассоциированных с шизофренией, могут включать NRG1, ErbB4, CPLX1, TPH1, TPH2, NRXN1, GSK3A, BDNF, DISC1, GSK3B и их комбинации.
Примеры белков, вовлеченных в подавление опухоли, могут включать, например, ATM (мутированный, атаксия-телеангиэктазия (ataxia telangiectasia mutated)), ATR (атаксия-телеангиэктазия и Rad3-родственный (ataxia telangiectasia and Rad3 related)), EGFR (рецептор эпидермального фактора роста (epidermal growth factor receptor)), ERBB2 (гомолог 2 v-erb-b2 эритробластического лейкоза вирусного онкогена (v-erb-b2 erythroblastic leukemia viral oncogene homolog 2)), ERBB3 (гомолог 3 v-erb-b2 эритробластического лейкоза вирусного онкогена (v-erb-b2 erythroblastic leukemia viral oncogene homolog 3)), ERBB4 (гомолог 4 v-erb-b2 эритробластического лейкоза вирусного онкогена (v-erb-b2 erythroblastic leukemia viral oncogene homolog 4)), Notch 1, Notch2, Notch 3 или Notch 4.
Примеры белков, ассоциированных с нарушением, связанным с секретазой, могут включать, например, PSENEN (presenilin enhancer 2 homolog (C. elegans)), CTSB (катепсин B (cathepsin B)), PSEN1 (пресенилин 1 (presenilin 1)), APP (белок-предшественник бета-амилоида (A4) (amyloid beta (A4) precursor protein)), APH1B (anterior pharynx defective 1 homolog B (C. elegans)), PSEN2 (пресенилин 2 (болезнь Альцгеймера 4) (presenilin 2 (Alzheimer disease 4)) или BACE1 (APP-расщепляющий фермент 1 по бета-сайту (beta-site APP-cleaving enzyme 1)).
Примеры белков, ассоциированных с амиотрофическим латеральным склерозом, могут включать, например, SOD1 (супероксиддисмутазу 1 (superoxide dismutase 1)), ALS2 (белок, ассоциированный с амиотрофическим латеральным склерозом 2 (amyotrophic lateral sclerosis 2)), FUS (РНК-связывающий белок FUS (fused in sarcoma)), TARDBP (TAR-ДНК связывающий белок (TAR DNA binding protein)), VAGFA (фактор роста эндотелия сосудов A (vascular endothelial growth factor A)), VAGFB (фактор роста эндотелия сосудов B (vascular endothelial growth factor B)) и VAGFC (фактор роста эндотелия сосудов C (vascular endothelial growth factor C)) и любую их комбинацию.
Примеры белков, ассоциированных с прионными болезнями, могут включать SOD1 (супероксиддисмутазу 1), ALS2 (белок, ассоциированный с амиотрофическим латеральным склерозом 2 (amyotrophic lateral sclerosis 2)), FUS (РНК-связывающий белок FUS (fused in sarcoma)), TARDBP (TAR-ДНК связывающий белок (TAR DNA binding protein)), VAGFA (фактор роста эндотелия сосудов A (vascular endothelial growth factor A)), VAGFB (фактор роста эндотелия сосудов B (vascular endothelial growth factor B)) и VAGFC (фактор роста эндотелия сосудов C (vascular endothelial growth factor C)) и любую их комбинацию.
Примеры белков, связанных с нейродегенеративными состояниями при прионных болезнях, могут включать, например, A2M (альфа-2-макроглобулин (Alpha-2-Macroglobulin)), AATF (фактор транскрипции, противодействующий апоптозу (Apoptosis antagonizing transcription factor)), ACPP (простатоспецифическую кислую фосфатазу (Acid phosphatase prostate)), ACTA2 (альфа-актин 2 гладкой мускулатуры аорты (Actin alpha 2 smooth muscle aorta)), ADAM22 (ADAM, металлопептидазный домен (ADAM metallopeptidase domain)), ADORA3 (аденозиновый рецептор A3 типа (Adenosine A3 receptor)) или ADRA1D (альфа-1D адренергический рецептор для альфа-1D адренорецептора (Alpha-1D adrenergic receptor for Alpha-1D adrenoreceptor)).
Примеры белков, ассоциированных с иммунодефицитом, могут включать, например, A2M [альфа-2-макроглобулин (alpha-2-macroglobulin)]; AANAT [арилалкиламин-N-ацетилтрансферазу (arylalkylamine N-acetyltransferase)]; ABCA1 [АТФ-связывающую кассету, подсемейство A (ABC1), представитель 1 (ATP-binding cassette, sub-family A (ABC1), member 1)]; ABCA2 [АТФ-связывающую кассету, подсемейство A (ABC1), представитель 2 (ATP-binding cassette, sub-family A (ABC1), member 2)] или ABCA3 [АТФ-связывающую кассету, подсемейство A (ABC1), представитель 3 (ATP-binding cassette, sub-family A (ABC1), member 3)].
Примеры белков, ассоциированных с нарушениями, связанными с тринуклеотидным повтором, включают, например, AR (андрогеновый рецептор (androgen receptor)), FMR1 (белок 1, ассоциированный с умственной отсталостью, связанной с ломкой X-хромосомой (fragile X mental retardation 1)), HTT (хантигтин (huntingtin)) или DMPK (протеинкиназу, ассоциированную с мышечной дистрофией (dystrophia myotonica-protein kinase)), FXN (фратаксин (frataxin)), ATXN2 (атаксин 2 (ataxin 2)).
Примеры белков, ассоциированных с нарушениями передачи нервных импульсов включают, например, SST (соматостатин (somatostatin)), NOS1 (синтазу оксида азота 1 (нейрональную) (nitric oxide synthase 1 (neuronal)), ADRA2A (адренергический, альфа-2A-, рецептор (adrenergic, alpha-2A-, receptor)), ADRA2C (адренергический, альфа-2C-, рецептор (adrenergic, alpha-2C-, receptor)), TACR1 (тахикининовый рецептор 1 (tachykinin receptor 1)) или HTR2c (5-гидрокситриптаминовый (серотониновый) рецептор 2C (5-hydroxytryptamine (serotonin) receptor 2C)).
Примеры последовательностей, ассоциированных с неврологическим развитием, включают, например, A2BP1 [атаксин 2-связывающий белок 1 (ataxin 2-binding protein 1)], AADAT [аминоадипатаминотрансферазу (aminoadipate aminotransferase)], AANAT [арилалкиламин-N-ацетилтрансферазу (arylalkylamine N-acetyltransferase)], ABAT [4-аминобутиратаминтрансферазу (4-aminobutyrate aminotransferase)], ABCA1 [АТФ-связывающую кассету, подсемейство A (ABC1), представитель 1 (ATP-binding cassette, sub-family A (ABC1), member 1)] или ABCA13 [АТФ-связывающую кассету, подсемейство A (ABC1), представитель 13 (ATP-binding cassette, sub-family A (ABC1), member 13)].
Дополнительные примеры предпочтительных состояний, которые подлежат лечению с помощью данной системы, включают те, которые могут быть выбраны из синдрома Айкарди-Гутьереса; болезни Александера; синдрома Аллана-Херндона-Дадли; связанных с геном POLG нарушений; альфа-маннозидоза (II и III тип); синдрома Альстрема; синдрома Ангельмана; атаксии-телеангиэктазии; нейронного высоковидного липофусциноза; бета-талассемии; двусторонней атрофии зрительного нерва и (инфантильной) атрофии зрительного нерва 1 типа; ретинобластомы (двусторонней); болезни Канавана; церебро-окуло-фацио-скелетного синдрома 1 [COFS1]; церебротендинального ксантоматоза; синдрома Корнелии де Ланге; связанных с геном MAPT нарушений; наследственных прионных болезней; синдрома Драве; семейной болезни Альцгеймера с ранним началом; атаксии Фридрейха [FRDA]; синдрома Фринса; фукозидоза; врожденной мышечной дистрофии Фукуямы; галактосиалидоза; болезни Гоше; органической ацидемии; гемофагоцитарного лимфогистиоцитоза; синдрома прогерии Гетчинсона-Гилфорда; муколипидоза II; инфантильной болезни накопления свободной сиаловой кислоты; ассоциированной с геном PLA2G6 нейродегенерации; синдрома Джервелла-Ланге-Нильсена; узелкового врожденного буллезного эпидермолиза; болезни Гентингтона; болезни Краббе (инфантильной); ассоциированного с митохондриальной ДНК синдрома Ли и NARP; синдрома Леша-Найхана; ассоциированной с геном LIS1 лиссэнцефалии; синдрома Лоу; болезни "кленового сиропа"; синдрома дупликации MECP2; связанных с геном ATP7A нарушений обмена меди; связанной с геном LAMA2 мышечной дистрофии; недостаточности арилсульфатазы А; мукополисахаридоза I, II или III типов; связанных с биогенезом пероксисом нарушений, спектра заболеваний по типу синдрома Цельвегера; нарушений по типу нейродегенерации с накоплением железа в головном мозге; недостаточности кислой сфингомиелиназы; болезни Ниманна-Пика C типа; глициновой энцефалопатии; связанных с геном ARX нарушений; нарушений орнитинового цикла; связанного с геном COL1A1/2 несовершенного остеогенеза; синдромов удаления митохондриальной ДНК; связанных с геном PLP1 нарушений; синдрома Перри; синдрома Фелана-МакДермида; болезни накопления гликогена II типа (болезни Помпе) (инфантильной); связанных с геном MAPT нарушений; связанных с геном MECP2 нарушений; эпифизарной точечной хондродисплазии 1 типа костей верхних конечностей или бедренной кости; синдрома Робертса; болезни Сандхоффа; болезни Шиндлера 1 типа; аденозиндезаминазной недостаточности; синдрома Смита-Лемли-Опитца; спинальной мышечной атрофии; спинально-церебеллярной атаксии с возникновением в младенческом возрасте; недостаточности гексозаминидазы А; танатофорной дисплазии 1 типа; связанных с геном коллагена VI типа нарушений; синдрома Ашера I типа; врожденной мышечной дистрофии; синдрома Вольфа-Хиршхорна; недостаточности лизосомной кислой липазы и пигментной ксеродермы.
Как будет понятно, предусматривается, что систему по настоящему изобретению можно использовать для целенаправленного воздействия на любую представляющую интерес полинуклеотидную последовательность. Некоторые состояния или заболевания, которые можно эффективно лечить с использованием системы по настоящему изобретению, включены в таблицы выше, и примеры известных на данный момент генов, ассоциированных с такими состояниями, также предоставлены в них. Тем не менее, гены, приведенные в качестве примеров, не являются исчерпывающими.
ПРИМЕРЫ
Следующие примеры приведены с целью иллюстрации различных вариантов осуществления настоящего изобретения и не предназначены для ограничения настоящего изобретения каким-либо образом. Данные примеры совместно со способами, описанными в данном документе, в настоящее время отражают предпочтительные варианты осуществления, являются иллюстративными и не предназначены для ограничения объема настоящего изобретения. Изменения в данном документе и другие применения, которые охватываются сущностью настоящего изобретения, как определено объемом формулы изобретения, будут очевидны специалистам в данной области.
Пример 1: активность комплекса CRISPR в ядре эукариотической клетки
Примером системы CRISPR II типа является локус CRISPR II типа из Streptococcus pyogenes SF370, который содержит кластер из 4 генов Cas9, Cas1, Cas2 и Csn1, а также два некодирующих элемента РНК, tracrRNA и характерный массив повторяющихся последовательностей (прямых повторов), чередующихся с короткими фрагментами неповторяющихся последовательностей (спейсерами, примерно 30 п.о. каждый). В этой системе целенаправленный двухцепочечный разрыв (DSB) ДНК образовывается в ходе четырех последовательных стадий (фигура 2A). Во-первых, две некодирующие РНК, массив pre-crRNA и tracrRNA транскрибируются с локуса CRISPR. Во-вторых, tracrRNA гибридизируется с прямыми повторами pre-crRNA, которая затем процессируется в зрелые crRNA, содержащие индивидуальные спейсерные последовательности. В-третьих, комплекс зрелая crRNA:tracrRNA направляет Cas9 к ДНК-мишени, состоящей из протоспейсера и соответствующего PAM, посредством образования гетеродуплекса между спейсерным участком crRNA и протоспейсерной ДНК. И наконец, Cas9 опосредует расщепление целевой ДНК выше PAM с образованием DSB внутри протоспейсера (фигура 2A). В данном примере описывается иллюстративный способ приспособления этой РНК-программируемой нуклеазной системы к управлению активностью комплекса CRISPR в ядрах эукариотических клеток.
Клеточная культура и трансфекция
Линию клеток почки эмбриона человека (HEK) 293FT (Life Technologies) поддерживали в среде Игла в модификации Дульбекко (DMEM), дополненной 10% фетальной бычьей сывороткой (HyClone), 2 мМ GlutaMAX (Life Technologies), 100 ЕД/мл пенициллина и 100 мкг/мл стрептомицина, при 37ºC с инкубированием при 5% CO2. Клеточную линию нейробластомы мышей (N2A) (ATCC) поддерживали в DMEM, дополненной 5% фетальной бычьей сывороткой (HyClone), 2 мМ GlutaMAX (Life Technologies), 100 ед/мл пенициллина и 100 мкг/мл стрептомицина, при 37ºC и 5% CO2.
Клетки HEK 293FT или N2A высевали в 24-луночные планшеты (Corning) за один день до трансфекции с плотностью 200000 клеток на лунку. Клетки трансфицировали с применением Lipofectamine 2000 (Life Technologies), следуя рекомендованному производителем протоколу. Для каждой лунки 24-луночного планшета использовали в общей сложности 800 нг плазмид.
Анализ с помощью Surveyor и анализ с помощью секвенирования на предмет наличия модификации генома
Клетки HEK 293FT или N2A трансфицировали плазмидной ДНК, как описано выше. После трансфекции клетки инкубировали при 37ºC в течение 72 часов перед экстракцией геномной ДНК. Геномную ДНК экстрагировали с помощью набора QuickExtract DNA extraction kit (Epicentre), следуя протоколу производителя. Вкратце, клетки ресуспендировали в растворе QuickExtract и инкубировали при 65ºC в течение 15 минут и при 98ºC в течение 10 минут. Экстрагированную геномную ДНК подвергали немедленной обработке или хранили при –20ºC.
Геномный участок, окружающий целевой сайт CRISPR, для каждого гена подвергали ПЦР амплификации и продукты очищали с использованием колонки QiaQuick Spin Column (Qiagen), следуя протоколу производителя. В общей сложности 400 нг очищенных ПЦР-продуктов смешивали с 2 мкл 10X ПЦР-буфера для Taq-полимеразы (Enzymatics) и водой сверхвысокой чистоты до конечного объема 20 мкл и подвергали процессу повторной гибридизации для обеспечения образования гетеродуплекса: 95ºC в течение 10 мин., линейное снижение температуры с 95ºC до 85ºC со скоростью 2ºC/с, с 85ºC до 25ºC со скоростью 0,25ºC/с и с выдерживанием при 25ºC в течение 1 минуты. После повторного отжига продукты обрабатывали нуклеазой Surveyor и энхансером S Surveyor (Transgenomics), следуя рекомендованному производителем протоколу, и анализировали в 4-20% полиакриламидных гелях Novex TBE (Life Technologies). Гели окрашивали красителем ДНК SYBR Gold (Life Technologies) в течение 30 минут и получали изображение с помощью системы обработки изображений Gel Doc gel imaging system (Bio-rad). Количественный анализ основывался на относительных интенсивностях полос в качестве единицы измерения фракции расщепленной ДНК. На фигуре 7 представлена схематическая иллюстрация данного анализа с помощью Surveyor.
Анализ полиморфизма длины рестрикционных фрагментов для обнаружения гомологичной рекомбинации.
Клетки HEK 293FT и N2A трансфицировали плазмидной ДНК и инкубировали при 37°C в течение 72 часов перед экстракцией геномной ДНК, как описано выше. Целевой геномный участок подвергали ПЦР амплификации с использованием праймеров за пределами гомологичных плеч матрицы для гомологичной рекомбинации (HR). ПЦР-продукты разделяли в 1% агарозном геле и экстрагировали с помощью набора MinElute GelExtraction Kit (Qiagen). Очищенные продукты расщепляли с помощью HindIII (Fermentas) и анализировали в 6% полиакриламидном геле Novex TBE (Life Technologies).
Прогнозирование и анализ вторичной структуры РНК
Прогнозирование вторичной структуры РНК осуществляли с использованием доступного в режиме онлайн веб-сервера RNAfold, разработанного в Институте теоретической химии при Венском университете, с использованием алгоритма прогнозирования структуры на основе центроидного метода (см., например, A.R. Gruber et al., 2008, Cell 106(1): 23-24; и PA Carr and GM Church, 2009, Nature Biotechnology 27(12): 1151-62).
Очистка РНК
Клетки HEK 293FT поддерживали и трансфицировали, как указано выше. Клетки собирали путем трипсинизации с последующим промыванием в фосфатно-солевом буфере (PBS). Общую клеточную РНК экстрагировали с помощью реагента TRI (Sigma), следуя протоколу производителя. Выполняли количественный анализ общей экстрагированной РНК с использованием Naonodrop (Thermo Scientific) и данные нормализовали к такой же концентрации.
Анализ экспрессии crRNA и tracrRNA в клетках млекопитающих с помощью нозерн-блоттинга
РНК смешивали с равными объемами 2X загрузочного буфера (Ambion), нагревали до 95°C в течение 5 мин., охлаждали на льду в течение 1 мин., а затем загружали в 8% денатурирующие полиакриламидные гели (SequaGel, National Diagnostics) после предварительного прогона геля в течение по меньшей мере 30 минут. Образцы подвергали электрофорезу в течение 1,5 часа при предельной мощности 40 Вт. После этого РНК переносили на мембрану Hybond N+ (GE Healthcare) при силе тока 300 мA в устройстве полусухого переноса (Bio-rad) при комнатной температуре в течение 1,5 часа. РНК сшивали с мембраной с использованием кнопки "автоматического сшивания" на приборе для автоматического сшивания с помощью ультрафиолета Stratagene UV Crosslinker the Stratalinker (Stratagene). Мембрану подвергали предварительной гибридизации в буфере для гибридизации ULTRAhyb-Oligo Hybridization Buffer (Ambion) в течение 30 мин с вращением при 42°C, а затем добавляли зонды и проводили гибридизацию в течение ночи. Зонды заказывали у IDT и метили [гамма-32P] ATP (Perkin Elmer) с использованием полинуклеотид-киназы T4 (New England Biolabs). Мембрану промывали один раз предварительно подогретым (42°C) 2xSSC, 0,5% SDS в течение 1 мин. с последующими двумя промывками по 30 минут при 42°C. Мембрану экспонировали на люминесцентном экране в течение одного часа или в течение ночи при комнатной температуре, а затем сканировали с использованием устройства для формирования изображения на люминесцентном фосфорном покрытии (Typhoon).
Конструирование и оценка бактериальной системы CRISPR
Элементы локуса CRISPR, в том числе tracrRNA, Cas9 и лидерную последовательность, подвергали ПЦР амплификации из геномной ДНК Streptococcus pyogenes SF370 с фланкирующими гомологичными плечами для сборки по методу Гибсона. Два сайта BsaI типа IIS вводили между двумя прямыми повторами для обеспечения вставки спейсеров (фигура 8). ПЦР-продукты клонировали в расщепленный с помощью EcoRV pACYC184 ниже промотора tet с использованием мастер-микса для сборки по методу Гибсона Gibson Assembly Master Mix (NEB). Другие эндогенные элементы системы CRISPR не включали, за исключением последних 50 п.о. в Csn2. Олигонуклеотиды (Integrated DNA Technology), кодирующие спейсеры с комплементарными липкими концами клонировали в расщепленный с помощью BsaI вектор pDC000 (NEB), а затем лигировали с использованием лигазы T7 (Enzymatics) с получением плазмид pCRISPR. Контрольные плазмиды, содержащие спейсеры, с экспрессией PAM в клетках млекопитающих (экспрессирующие конструкции, проиллюстрированные на фигуре 6A, с функциональностью, которая определена по результатам анализа с использованием Surveyor, показанным на фигуре 6B). Сайты инициации транскрипции обозначены как +1, а также указаны терминатор транскрипции и последовательность, гибридизирующаяся с зондом при нозерн-блоттинге. Экспрессию подвергнутой процессингу tracrRNA также подтверждали с помощью нозерн-блоттинга. На фигуре 6C показаны результаты анализа с помощью нозерн-блоттинга общей РНК, экстрагированной из клеток 293FT, трансфицированных экспрессирующими конструкциями U6, несущими длинную или короткую tracrRNA, а также SpCas9 и DR-EMX1(1)-DR. Левая и правая секции получены с клетками 293FT, трансфицированными без SpRNase III или с таковой, соответственно. U6 являются показателем для контроля загрузки при блоттинге с зондом, нацеленным на малую ядерную РНК (snRNA) U6 человека. Трансфекция экспрессирующей конструкции с короткой tracrRNA приводит к избыточным уровням подвергшейся процессингу формы tracrRNA (~75 п.о.). Очень низкие количества длинных tracrRNA обнаруживали при нозерн-блоттинге.
Для стимуляции точной инициации транскрипции промотор U6 на основе РНК-полимеразы III выбирали для управления экспрессией tracrRNA (фигура 2C). Подобным образом, конструкцию на основе промотора U6 разрабатывали для экспрессии массива pre-crRNA, состоящего из одного спейсера, фланкированного двумя прямыми повторами (DR, также включены в выражение “парные tracr-последовательности”; фигура 2C). Исходный спейсер был разработан для целенаправленного воздействия на целевой сайт из 33 пар оснований (п.о.) (протоспейсер из 30 п.о., а также последовательность мотива CRISPR (PAM) из 3 п.о., соответствующая мотиву узнавания NGG у Cas9) в локусе EMX1 человека (фигура 2C), ключевом гене в развитии коры головного мозга.
Клетки HEK 293FT трансфицировали комбинациями компонентов CRISPR для того, чтобы определить, возможно ли при гетерологичной экспрессии системы CRISPR (SpCas9, SpRNase III, tracrRNA и pre-crRNA) в клетках млекопитающих достичь целенаправленного расщепления хромосом млекопитающего. Поскольку DSB в ядрах млекопитающих подвергаются частичной репарации с помощью пути негомологичного соединения концов (NHEJ), который приводит к формированию вставок/делеций, анализ с помощью SURVEYOR использовали для выявления возможной активности для расщепления в целевом локусе EMX1 (фигура 7) (см., например, Guschin et al., 2010, Methods Mol Biol 649: 247). Котрансфекция всех четырех компонентов CRISPR была способна индуцировать расщепления в протоспейсере на уровне до 5,0% (см. фигуру 2D). Котрансфекция всех компонентов CRISPR, за исключением SpRNase III, также индуцировала образование вставок/делеций в протоспейсере на уровне до 4,7%, что указывало на то, что могут существовать эндогенные РНКазы млекопитающих, которые способны помогать созреванию crRNA, такие как, например, родственные ферменты Dicer и Drosha. Удаление любого из трех остальных компонентов ликвидировало активность системы CRISPR для расщепления генома (фигура 2D). Секвенирование по Сэнгеру ампликонов, содержащих целевой локус, подтверждало активность для расщепления: на 43 подвергшихся секвенированию клонов было обнаружено 5 мутированных аллелей (11,6%). В подобных экспериментах с использованием ряда направляющих последовательностей процентные значения содержания вставок/делеций составляли до 29% (см. фигуры 3-6, 10 и 11). Эти результаты определяют трехкомпонентную систему для эффективной опосредованной CRISPR модификации генома в клетках млекопитающих. Для оптимизации эффективности расщепления заявители также определяли, влияют ли различные изоформы tracrRNA на эффективность расщепления, и обнаружили, что в этой иллюстративной системе только короткая (89 п.о.) форма транскрипта была способна опосредовать расщепление локуса генома EMX1 человека (фигура 6B).
На фигуре 12 представлен дополнительный анализ процессинга crRNA в клетках млекопитающих с помощью нозерн-блоттинга. На фигуре 12A показано схематическое изображение вектора экспрессии для одного спейсера, фланкированного двумя прямыми повторами (DR-EMX1(1)-DR). Спейсер из 30 п.о., нацеленный на протоспейсер 1 локуса EMX1 человека (см. фигуру 6), и последовательности прямых повторов показаны в последовательности внизу фигуры 12A. Линия указывает на участок, обратно комплементарную последовательность которого использовали для создания зондов для нозерн-блоттинга для обнаружения crRNA EMX1(1). На фигуре 12B показаны результаты анализа с помощью нозерн-блоттинга общей РНК, экстрагированной из клеток 293FT, трансфицированных экспрессирующими конструкциями U6, несущими DR-EMX1(1)-DR. Левая и правая секции получены с клетками 293FT, трансфицированными без SpRNase III или с таковой, соответственно. DR-EMX1(1)-DR подвергался процессингу в зрелые crRNA только в присутствии SpCas9 и короткой tracrRNA и не зависел от присутствия SpRNase III. Зрелая crRNA, обнаруженная в общей РНК трансфицированных 293FT, имела длину ~33 п.о. и была короче, чем зрелая crRNA из S. pyogenes длиной 39-42 п.о. Данные результаты демонстрируют, что систему CRISPR можно перенести в эукариотические клетки и перепрограммировать для облегчения расщепления эндогенных целевых полинуклеотидов млекопитающих.
На фигуре 2 показана бактериальная система CRISPR, описанная в этом примере. На фигуре 2A показано схематическое изображение локуса 1 CRISPR из Streptococcus pyogenes SF370 и предполагаемый механизм опосредованного CRISPR расщепления ДНК с помощью этой системы. Зрелая crRNA, подвергшаяся процессингу из массива прямых повторов-спейсеров, направляет Cas9 к мишеням в геноме, состоящим из комплементарных протоспейсеров и мотива, смежного с протоспейсером (PAM). При спаривании оснований мишень-спейсер Cas9 опосредует двухцепочечный разрыв в целевой ДНК. На фигуре 2B показано конструирование Cas9 S. pyogenes (SpCas9) и RNase III (SpRNase III) с сигналами ядерной локализации (NLS) для обеспечения импорта в ядро млекопитающих. На фигуре 2C показана экспрессия SpCas9 и SpRNase III у млекопитающих, управляемая конститутивным промотором EF1a, и массива tracrRNA и pre-crRNA (DR-спейсер-DR), управляемая промотором U6 РНК-полимеразы 3 для стимуляции точной инициации и терминации транскрипции. Протоспейсер из локуса EMX1 человека с удовлетворительной последовательностью PAM использовали в качестве спейсера в массиве pre-crRNA. На фигуре 2D показан анализ с помощью нуклеазы Surveyor в отношении опосредованных SpCas9 минорных вставок и делеций. SpCas9 экспрессировался с SpRNase III, tracrRNA и массивом pre-crRNA, несущим целевой спейсер для EMX1, и без таковых. На фигуре 2E показано схематическое изображение спаривания оснований между целевым локусом и нацеленной на EMX1 crRNA, а также иллюстративная хроматограмма, на которой показана микроделеция, смежная по отношению к сайту расщепления SpCas9. На фигуре 2F показаны мутированные аллели, идентифицированные в результате анализа секвенирования 43 клональных ампликонов, показывающие разнообразие микровставок и микроделеций. Штрихами указаны удаленные основания, а невыровненные или несовпадающие основания указывают на вставки или мутации. Масштабная метка = 10 мкм.
Для дальнейшего упрощения трехкомпонентной системы адаптировали гибридную структуру химерная crRNA-tracrRNA, в которой зрелая crRNA (содержащую направляющую последовательность) может быть слита с частичной tracrRNA через структуру по типу стебель-петля для имитации естественного дуплекса crRNA:tracrRNA. Для повышения эффективности совместной доставки создавали бицистронный вектор экспрессии для управления коэкспрессией химерной РНК и SpCas9 в трансфицированных клетках. Параллельно, бицистронные векторы использовали для экспрессии pre-crRNA (DR-направляющая последовательность-DR) с SpCas9, чтобы индуцировать процессинг в crRNA с участием отдельно экспрессируемой tracrRNA (сравнение на фигуре 11B: верхняя часть и нижняя часть). На фигуре 8 представлены схематические иллюстрации бицистронных векторов экспрессии для массива pre-crRNA (фигура 8A) или химерных crRNA (представлено короткой линией ниже сайта встраивания направляющей последовательности и выше промотора EF1α на фигуре 8B) с hSpCas9, показывающие положение различных элементов и точки встраивания направляющей последовательности. В расширенной последовательности вокруг положения сайта встраивания направляющей последовательности на фигуре 8B также показана частичная последовательность DR (GTTTTAGAGCTA) (SEQ ID NO: 11) и частичная последовательность tracrRNA (TAGCAAGTTAAAATAAGGCTAGTCCGTTTTT) (SEQ ID NO: 12). Направляющие последовательности можно встроить между сайтами BbsI с использованием гибридизированных олигонуклеотидов. Структуры последовательностей для олигонуклеотидов показаны ниже схематических иллюстраций на фигуре 8 с указанными подходящими адаптерами лигирования. WPRE представляет собой посттранскрипционный регуляторный элемент вируса гепатита сурков. Эффективность опосредованного химерной РНК расщепления исследовали путем целенаправленного воздействия на тот же локус EMX1, описанный выше. С использованием как анализа с помощью Surveyor, так и секвенирования ампликонов по Сэнгеру заявители подтвердили, что химерная структура РНК облегчает расщепление локуса EMX1 человека со степенью модификации примерно 4,7% (фигура 3).
Генерализованность опосредованного CRISPR расщепления в эукариотических клетках исследовали путем целенаправленного воздействия на дополнительные локусы генома как в клетках человека, так и мыши путем разработки химерных РНК, целенаправленно воздействующих на множество сайтов в EMX1 и PVALB человека, а также на локусы Th мыши. На фигуре 13 показан выбор некоторых дополнительных служащих в качестве мишени протоспейсеров в локусах PVALB человека (фигура 13A) и Th мыши (фигура 13B). Приведены схематические изображения локусов генов и положения трех протоспейсеров в последнем экзоне каждого из них. Подчеркнутые последовательности включают 30 п.о. последовательности протоспейсера и 3 п.о. на 3’ конце, соответствующие последовательностям PAM. Протоспейсеры на смысловой и антисмысловой нитях указаны выше и ниже последовательностей ДНК, соответственно. Степень модификации 6,3% и 0,75% достигали для локусов PVALB человека и Th мыши, соответственно, демонстрируя широкую применимость системы CRISPR при модификации различных локусов у нескольких организмов (фигура 5). Хотя при использовании химерных конструкций расщепление обнаруживали только с одним из трех спейсеров для каждого локуса, все целевые последовательности расщеплялись с эффективностью получения вставок/делеций, достигающей 27%, при использовании схемы с коэкспрессируемой pre-crRNA (фигуры 6 и 13).
На фигуре 11 представлена дополнительная иллюстрация того, что SpCas9 можно перепрограммировать для целенаправленного воздействия на несколько локусов генома в клетках млекопитающих. На фигуре 11A представлено схематическое изображение локуса EMX1 человека, на котором показано положение пяти протоспейсеров, указанных с помощью подчеркнутых последовательностей. На фигуре 11B представлено схематическое изображение комплекса pre-crRNA/trcrRNA, на котором показана гибридизация между участком прямого повтора в pre-crRNA и tracrRNA (вверху), и схематическое изображение структуры химерной РНК, содержащей направляющую последовательность из 20 п.о., и парную tracr-последовательность, и tracr-последовательность, состоящие из неполного прямого повтора и последовательностей tracrRNA, гибридизированных в "шпилечную" структуру (внизу). Результаты анализа с помощью Surveyor со сравнением эффективности опосредованного Cas9 расщепления в пяти протоспейсерах в локусе EMX1 человека показаны на фигуре 11C. Целенаправленное воздействие на каждый протоспейсер осуществляли либо с использованием подвергнутого процессингу комплекса pre-crRNA/tracrRNA (crRNA), либо с использованием химерной РНК (chiRNA).
Поскольку вторичная структура РНК может быть важной для межмолекулярных взаимодействий, алгоритм предсказания структуры на основе минимальной свободной энергии и ансамбля взвешенных структур по Больцману использовали для сравнения предполагаемой вторичной структуры всех направляющих последовательностей, используемых в эксперименте с целенаправленным воздействием на геном (фигура 3B) (см., например, Gruber et al., 2008, Nucleic Acids Research, 36: W70). Анализ выявил, что в большинстве случаев эффективные направляющие последовательности в контексте химерной crRNA, по сути, не содержали мотивов вторичной структуры, тогда как неэффективные направляющие последовательности с большей вероятностью образовывали внутренние вторичные структуры, которые могут препятствовать спариванию оснований с ДНК целевого протоспейсера. Следовательно, возможно, что вариабельность во вторичной структуре спейсера может оказывать воздействие на эффективность опосредованной CRISPR интерференции при использовании химерной crRNA.
Структуры дополнительных векторов для SpCas9 показаны на фигуре 22, на которой показаны отдельные векторы экспрессии, включающие промотор U6, сцепленный с сайтом встраивания для направляющего олигонуклеотида, и промотор Cbh, сцепленный с кодирующей последовательностью SpCas9. Вектор, показанный на фигуре 22b, включает кодирующую последовательность tracrRNA, сцепленную с промотором H1.
При анализе у бактерий все спейсеры способствовали эффективной CRISPR-интерференции (фигура 3C). Эти результаты указывают на то, что могут существовать дополнительные факторы, влияющие на эффективность активности CRISPR в клетках млекопитающих.
Для исследования специфичности опосредованного CRISPR расщепления эффект однонуклеотидных мутаций в направляющей последовательности в отношении расщепления протоспейсера в геноме млекопитающих анализировали с использованием ряда целенаправленно воздействующих на EMX химерных crRNA с единичными точковыми мутациями (фигура 3A). На фигуре 3B показаны результаты анализа с помощью нуклеазы Surveyor со сравнением эффективности расщепления Cas9 при спаривании с различными мутантными химерными РНК. Несовпадение одного основания в участке вплоть до 12 п.о. с 5’ в PAM, по сути, прекращало расщепление генома SpCas9, тогда как спейсеры с мутациями в положениях, расположенных в более отдаленных положениях выше относительно хода транскрипции сохраняли активность в отношении исходного протоспейсера-мишени (фигура 3B). В дополнение к PAM, SpCas9 характеризуется специфичностью в отношении одного основания в последних 12 п.о. спейсера. Кроме того, CRISPR способен опосредовать расщепление генома столь же эффективно, как и пара нуклеаз TALE (TALEN), целенаправленно воздействующих на тот же протоспейсер EMX1. На фигуре 3C представлено схематическое изображение, на котором показана структура TALEN, целенаправленно воздействующих на EMX1, и на фигуре 3D показано сравнение эффективности TALEN и Cas9 (n=3) при разгонке в геле продуктов, полученных в результате анализа с помощью Surveyor.
Установив набор компонентов для достижения опосредованного CRISPR редактирования генов в клетках млекопитающих посредством склонного к ошибкам механизма NHEJ, исследовали способность CRISPR к стимуляции гомологичной рекомбинации (HR), высокоточный путь репарации генов для создания точных редакционных изменений в геноме. SpCas9 дикого типа способен опосредовать сайт-специфические DSB, которые могут репарироваться как с помощью NHEJ, так и HR. Кроме того, замену аспартата на аланин (D10A) в каталитическом домене RuvC I в SpCas9 производили посредством методик генетической инженерии для превращения нуклеазы в никазу (SpCas9n; проиллюстрировано на фигуре 4A) (см., например, Sapranausaks et al., 2011, Nucleic Acids Research, 39: 9275; Gasiunas et al., 2012, Proc. Natl. Acad. Sci. USA, 109:E2579) так, чтобы надрезанная геномная ДНК подвергалась высокоточной репарации с участием гомологичной рекомбинации (HDR). Анализ с помощью Surveyor подтвердил, что SpCas9n не создает вставок/делеций в протоспейсере-мишени EMX1. Как показано на фигуре 4B, коэкспрессия целенаправленно воздействующей на EMX1 химерной crRNA с SpCas9 давала вставки/делеции в целевом сайте, тогда как коэкспрессия с SpCas9n - нет (n=3). Более того, секвенирование 327 ампликонов не обнаружило каких-либо вставок/делеций, индуцированных SpCas9n. Для исследования опосредованной CRISPR HR при совместной трансфекции клеток HEK 293FT химерной РНК, целенаправленно воздействующей на EMX1, hSpCas9 или hSpCas9n, выбирали тот же локус, также как и матрицу для HR для введения пары сайтов рестрикции (HindIII и NheI) возле протоспейсера. На фигуре 4C приведена схематическая иллюстрация стратегии HR с относительными положениями точек рекомбинации и последовательностей для гибридизации праймеров (стрелки). SpCas9 и SpCas9n действительно катализировали интеграцию матрицы HR в локус EMX1. ПЦР амплификация целевого участка с последующим рестрикционным расщеплением HindIII выявила продукты расщепления, соответствующие ожидаемым размерам фрагментов (стрелки на результатах анализа полиморфизма длин рестрикционных фрагментов с помощью гель-электрофореза, показанных на фигуре 4D), причем SpCas9 и SpCas9n опосредуют подобные уровни эффективности HR. Заявители дополнительно подтверждали HR с использованием секвенирования геномных ампликонов по Сэнгеру (фигура 4E). Эти результаты демонстрировали пригодность CRISPR для облегчения целенаправленной вставки генов в геном млекопитающего. С учетом специфичности к мишени в 14 п.о. (12 п.о. от спейсера и 2 п.о. от PAM) SpCas9 дикого типа доступность никазы может значительно снизить вероятность нецелевой модификации, поскольку одноцепочечные разрывы не являются субстратами для склонного к ошибкам пути NHEJ.
Экспрессирующие конструкции, имитирующие естественную архитектуру локусов CRISPR с собранными в массив спейсерами (фигура 2A), создавали для исследования возможности мультиплексного целенаправленного воздействия на последовательности. При использовании одного массива CRISPR, кодирующего пару спейсеров, нацеленных на EMX1 и PVALB, обнаруживали эффективное расщепление в обоих локусах (фигура 4F, на которой показаны как схематическая структура массива crRNA, так и блот, полученный после анализа с помощью Surveyor, показывающий эффективное опосредование расщепления). Также исследовали целенаправленную делецию геномных участков большего размера посредством одновременных DSB с использованием спейсеров против двух мишеней в EMX1, разделенных 119 п.о., и обнаруженная эффективность делеции составляла 1,6% (3 из 182 ампликонов; фигура 4G). Это демонстрирует, что система CRISPR может опосредовать мультиплексное редактирование в пределах одного генома.
Пример 2: модификации и альтернативы системы CRISPR
Возможность применения РНК для программирования специфичного к последовательности расщепления ДНК определяет новый класс инструментов для конструирования генома для разнообразных исследовательских и промышленных применений. Несколько аспектов системы CRISPR можно дополнительно улучшить для повышения эффективности и универсальности целенаправленного воздействия с помощью CRISPR. Оптимальная активность Cas9 может зависеть от доступности несвязанного Mg2+ на уровнях, которые превышают имеющиеся в ядре млекопитающего (см., например, Jinek et al., 2012, Science, 337:816), и предпочтение в отношении мотива NGG непосредственно ниже протоспейсера ограничивает способность к целенаправленному воздействию в среднем на каждые 12 п.о. в геноме человека (фигура 9, оценка как плюс-, так и минус-нитей в хромосомных последовательностях человека). Некоторые из этих затруднений можно преодолеть путем изучения разнообразия локусов CRISPR в микробном метагеноме (см., например, Makarova et al., 2011, Nat Rev Microbiol, 9:467). Другие локусы CRISPR можно переместить в микроокружение клетки млекопитающего с помощью способа, подобного описанному в примере 1. Например, на фигуре 10 показана адаптация системы CRISPR II типа из CRISPR 1 LMD-9 Streptococcus thermophilus для гетерологичной экспрессии в клетках млекопитающих, чтобы достичь опосредованного CRISPR редактирования генома. На фигуре 10A приведена схематическая иллюстрация CRISPR 1 из LMD-9 S. thermophilus. На фигуре 10B показана структура системы экспрессии для системы CRISPR S. thermophilus. Кодон-оптимизированный hStCas9 человека экспрессируется с помощью конститутивного промотора EF1α. Зрелые варианты tracrRNA и crRNA экспрессируются с помощью промотора U6 для стимуляции точной инициации транскрипции. Показаны последовательности из зрелых crRNA и tracrRNA. Одно основание, обозначенное буквой “a” в нижнем регистре в последовательности crRNA, использовали для удаления последовательности polyU, которая служит в качестве терминатора транскрипции РНК polIII. На фигуре 10C приведено схематическое изображение направляющих последовательностей, нацеленных на локус EMX1 человека. На фигуре 10D показаны результаты опосредованного hStCas9 расщепления в целевом локусе с использованием анализа с помощью Surveyor. РНК направляющих спейсеров 1 и 2 индуцировали 14% и 6,4%, соответственно. Статистический анализ активности для расщепления по биологическим копиям в этих двух протоспейсерных сайтах также приведен на фигуре 5. На фигуре 14 приведено схематическое изображение дополнительного протоспейсера и соответствующих последовательностей PAM, являющихся мишенями для системы CRISPR S. thermophilus, в локусе EMX1 человека. Последовательности двух протоспейсеров выделены, а их соответствующие последовательности PAM, удовлетворяющие мотиву NNAGAAW, обозначены путем подчеркивания в направлении 3’ относительно соответствующей выделенной последовательности. Оба протоспейсера нацелены на антисмысловую нить.
Пример 3: алгоритм выбора образцов целевой последовательности
Создали компьютерную программу для идентификации кандидатных целевых последовательностей CRISPR на обеих нитях вводимой последовательности ДНК на основе длины желаемой направляющей последовательности и мотива последовательности CRISPR (PAM) для определенного фермента CRISPR. Например, целевые сайты для Cas9 из S. pyogenes с последовательностями PAM NGG можно идентифицировать путем поиска в отношении 5’-Nx-NGG-3’ как на вводимой последовательности, так и на последовательности, обратно-комплементарной вводимой. Подобным образом, целевые сайты для Cas9 CRISPR1 S. thermophilus с последовательностью PAM NNAGAAW можно идентифицировать путем поиска в отношении 5’-Nx-NNAGAAW-3’ как на вводимой последовательности, так и на последовательности, обратно-комплементарной вводимой. Подобным образом, целевые сайты для Cas9 CRISPR3 S. thermophilus с последовательностью PAM NGGNG можно идентифицировать путем поиска в отношении 5’-Nx-NGGNG-3’ как на вводимой последовательности, так и на последовательности, обратно-комплементарной вводимой. Значение “x” в Nx может фиксироваться программой или может быть определено пользователем, как, например, 20.
Поскольку несколько случаев появления целевого сайта ДНК в геноме могут приводить к неспецифическому редактированию генома, после идентификации всех возможных сайтов программа профильтровывает последовательности, исходя из количества раз, когда они встречаются в соответствующем эталонном геноме. Для тех ферментов CRISPR, для которых специфичность к последовательности определяется "затравочной" последовательностью, такой как находящаяся в 11-12 п.о. в направлении 5’ от последовательности PAM, в том числе сама последовательность PAM, стадия фильтрования может основываться на "затравочной" последовательности. Следовательно, во избежание редактирования в дополнительных локусах генома результаты фильтруют, исходя из числа случаев обнаружения последовательности затравки:PAM в подходящем геноме. Пользователь может иметь возможность выбора длины затравочной последовательности. Пользователь также может иметь возможность определять число случаев обнаружения последовательности затравки:PAM в геноме применительно к прохождению фильтра. По умолчанию установлен скрининг в отношении уникальных последовательностей. Уровень фильтрования изменяют путем изменения как длины затравочной последовательности, так и числа случаев обнаружения последовательности в геноме. В качестве дополнения или альтернативы, программа может обеспечивать последовательность направляющей последовательности, комплементарную сообщенной(ым) целевой(ым) последовательности(ям) путем обеспечения последовательности, обратно комплементарной идентифицированной(ым) целевой(ым) последовательности(ям). Иллюстративная визуализация некоторых целевых сайтов в геноме человека представлена на фигуре 18.
Дальнейшие детали способов и алгоритмов для оптимизации выбора последовательности можно найти в заявке на патент США с серийным номером 61/064798 (номер дела у патентного поверенного 44790.11.2022; общая ссылка BI-2012/084); включенной в данный документ при помощи ссылки.
Пример 4: оценка нескольких гибридов химерная crRNA-tracrRNA
В данном примере описаны результаты, полученные для химерных РНК (chiRNA; содержащие направляющую последовательность, парную tracr-последовательность и tracr-последовательность в одном транскрипте), имеющих tracr-последовательности, которые включают фрагменты последовательности tracrRNA дикого типа с разной длиной. На фигуре 16a показано схематическое изображение бицистронного вектора экспрессии для химерной РНК и Cas9. Cas9 управляется промотором CBh, а химерная РНК управляется промотором U6. Химерная направляющая РНК состоит из направляющей последовательности (Ns) из 20 п.о., соединенной с tracr-последовательностью (проходящей от первого “U” в нижней нити к концу транскрипта), которая усечена в разных положениях, которые указаны. Направляющие и tracr-последовательности разделены парной tracr-последовательностью GUUUUAGAGCUA (SEQ ID NO: 13), за которой следует последовательность петли GAAA. Результаты анализов с помощью SURVEYOR в отношении опосредованных Cas9 вставок/делеций в локусах EMX1 и PVALB человека показаны на фигурах 16b и 16c, соответственно. Стрелки указывают на ожидаемые фрагменты, полученные в результате расщепления с помощью SURVEYOR. ChiRNA показаны с помощью обозначения их “+n”, а crRNA относится к гибридной РНК, в которой направляющие и tracr-последовательности экспрессируются в виде раздельных транскриптов. Количественный анализ этих результатов, выполненный в трех повторностях, проиллюстрирован с помощью гистограмм на фигурах 17a и 17b, соответствующих фигурам 16b и 16c, соответственно (“N.D.” означает отсутствие обнаруженных вставок/делеций). ID (идентификационные данные) протоспейсеров и их соответствующей мишени в геноме, последовательность протоспейсера, последовательность PAM и положение нити приведены в таблице D. Направляющие последовательности разработаны так, чтобы они были комплементарны полной последовательности протоспейсера в случае отдельных транскриптов в гибридной системе или только подчеркнутой части в случае химерных РНК.
Таблица D:
протоспейсера
в геноме
Дополнительные детали для оптимизации направляющих последовательностей можно найти в заявке на патент США с серийным номером 61/836127 (номер дела у патентного поверенного 44790.08.2022; общая ссылка BI-2013/004G); включенной в данный документ при помощи ссылки.
Первоначально целенаправленному воздействию подвергали три сайта в пределах локуса EMX1 в клетках HEK 293FT человека. Эффективность модификации генома каждой chiРНК оценивали с использованием анализа с помощью нуклеазы SURVEYOR, который позволяет обнаруживать мутации, возникающие в результате двухцепочечных разрывов (DSB) ДНК и их последующей репарации с помощью пути репарации повреждения ДНК за счет негомологичного соединения концов (NHEJ). В конструкциях, обозначенных chiRNA(+n), указывается, что нуклеотиды в количестве до +n нуклеотида tracrRNA дикого типа включены в химерную РНК-конструкцию, при этом для n используются значения 48, 54, 67 и 85. Химерные РНК, содержащие более длинные фрагменты tracrRNA дикого типа (chiRNA(+67) и chiRNA(+85)), опосредовали расщепление ДНК во всех трех целевых сайтах EMX1, причем chiRNA(+85), в частности, демонстрировал значительно более высокие уровни расщепления ДНК, чем соответствующие гибриды crRNA/tracrRNA, у которых направляющие и tracr-последовательности экспрессируются в отдельных транскриптах (фигуры 16b и 17a). Два сайта в локусе PVALB, которые не давали обнаруживаемого расщепления с использованием гибридной системы (направляющая последовательность и tracr-последовательность, экспрессируемые в виде отдельных транскриптов), также подвергались целенаправленному воздействию с использованием chiRNA. сhiRNA(+67) и chiRNA(+85) были способны опосредовать значительное расщепление в двух протоспейсерах в PVALB (фигуры 16c и 17b).
Для всех пяти мишеней в локусах EMX1 и PVALB наблюдали соответствующее повышение эффективности модификации генома с увеличением длины tracr-последовательности. Не вдаваясь в какую-либо теорию, вторичная структура, формируемая 3’ концом tracrRNA, может играть роль в увеличении скорости образования комплекса CRISPR.
Пример 5: разнообразие Cas9
Система CRISPR-Cas является адаптивным иммунным механизмом в отношении внедряющейся экзогенной ДНК, используемым разнообразными видами из числа бактерий и архей. Система CRISPR-Cas9 II типа состоит из набора генов, кодирующих белки, ответственные за “захват” чужеродной ДНК в локус CRISPR, а также из набора генов, кодирующих “выполнение” механизма расщепления ДНК; они включают ДНК-нуклеазу (Cas9), некодирующую транс-активирующую cr-RNA (tracrRNA) и массив полученных из чужеродной ДНК спейсеров, фланкированных прямыми повторами (crRNA). При созревании под действием Cas9 дуплекс tracRNA и crRNA направляет нуклеазу Cas9 к целевой последовательности ДНК, определенной спейсерными направляющими последовательностями, и опосредует двухцепочечные разрывы в ДНК вблизи короткого мотива последовательности в целевой ДНК, которые требуются для расщепления и являются специфичными для каждой системы CRISPR-Cas. Системы CRISPR-Cas II типа обнаруживаются повсеместно в царстве бактерий и являются очень разными по последовательности и размеру белка Cas9, последовательности прямого повтора tracrRNA и crRNA, организации этих элементов в геноме и требованиям к мотиву для целенаправленного расщепления. Один вид может иметь несколько отдельных систем CRISPR-Cas.
Заявители оценивали 207 предполагаемых Cas9 из видов бактерий, идентифицированных на основании гомологии последовательности с известными Cas9 и структурами, ортологичными известным субдоменам, в том числе домену эндонуклеазы HNH и доменам эндонуклеазы RuvC [информация от Eugene Koonin и Kira Makarova]. Филогенетический анализ, основанный на консервативности последовательности белка в этом наборе, выявил пять семейств Cas9, включая три группы больших Cas9 (~1400 аминокислот) и две малых Cas9 (~1100 аминокислот) (см. фигуры 19 и 20A-F).
Дополнительные детали по Cas9 и мутациям в ферменте Cas9 для превращения в никазу или ДНК-связывающий белок и их применению с измененной функциональностью можно найти в заявках на патент США с порядковыми номерами 61/836101 и 61/835936 (номера дел у патентного поверенного 44790.09.2022 и 4790.07.2022 и общие ссылки BI-2013/004E и BI-2013/004F, соответственно), включенных в данный документ при помощи ссылки.
Пример 6: ортологи Cas9
Заявители анализировали ортологи Cas9 для идентификации подходящих последовательностей PAM и соответствующей химерной направляющей РНК. Наличие расширенного набора PAM обеспечивает более широкое целенаправленное воздействие по геному, а также значительно повышает число уникальных целевых сайтов и обеспечивает возможность для идентификации новых Cas9 с повышенными уровнями специфичности в геноме.
Специфичность ортологов Cas9 можно оценить с помощью исследования способности каждого Cas9 допускать несовпадения между направляющей РНК и ДНК-мишенью. Например, специфичность SpCas9 была охарактеризована с помощью исследования эффекта мутаций в направляющей РНК в отношении эффективности расщепления. Создавали библиотеки направляющих РНК с одним или несколькими несовпадениями между направляющей последовательностью и целевой ДНК. Исходя из этих данных, целевые сайты для SpCas9 можно выбрать на основе следующих рекомендаций.
Для максимального повышения специфичности SpCas9 в отношении редактирования конкретного гена специалисту следует выбирать целевой сайт в пределах представляющего интерес локуса таким образом, чтобы в возможных "нецелевых" геномных последовательностях соблюдались следующие четыре ограничения: первое и главное, за ними не должен следовать PAM с любой из последовательностей 5’-NGG или NAG; второе, глобальное сходство их последовательности с целевой последовательностью должно быть сведено к минимуму; третье, максимальное число несовпадений должно находиться в пределах ближайшего к PAM участка в нецелевом сайте; наконец, максимальное число несовпадений должны быть последовательными или отделенными друг от друга менее чем четырьмя основаниями.
Подобные способы можно использовать для оценки специфичности других ортологов Cas9 и для установления критериев для выбора специфических целевых сайтов в геномах целевых видов. Как упоминалось ранее, филогенетический анализ, основанный на консервативности последовательности белка в этом наборе, выявил пять семейств Cas9, включая три группы больших Cas9 (~1400 аминокислот) и две малых Cas9 (~1100 аминокислот) (см. фигуры 19 и 20A-F). Дополнительные детали в отношении ортологов Cas можно найти в заявках на патент США с серийными номерами 61/836101 и 61/835936 (номера дел у патентного поверенного 44790.09.2022 и 4790.07.2022 и общие ссылки BI-2013/004E и BI-2013/004F, соответственно), включенных в данный документ при помощи ссылки.
Пример 7: конструирование растений (микроскопических водорослей) с использованием Cas9 для целенаправленного воздействия и манипуляции с генами растений
Способы доставки Cas9
Способ 1. Заявители доставляли Cas9 и направляющую РНК с применением вектора, экспрессирующего Cas9 под контролем конститутивного промотора, такого как промотор Hsp70A-Rbc S2 или бета-2-тубулиновый промотор.
Способ 2. Заявители доставляли Cas9 и полимеразу T7 с использованием векторов, экспрессирующих Cas9 и полимеразу T7 под контролем конститутивного промотора, такого как промотор Hsp70A-Rbc S2 или бета-2-тубулиновый промотор. Направляющая РНК будет доставляться с использованием вектора, содержащего промотор T7, управляющий экспрессией направляющей РНК.
Способ 3. заявители доставляли мРНК Cas9 и in vitro транскрибировали направляющую РНК в клетках водорослей. РНК можно транскрибировать in vitro. мРНК Cas9 будет состоять из кодирующего участка для Cas9, а также 3’UTR из Cop1, чтобы обеспечивать стабилизацию мРНК Cas9.
Для гомологичной рекомбинации заявители обеспечивали дополнительную матрицу для репарации с участием гомологичной рекомбинации.
Последовательность для кассеты, управляющей экспрессией Cas9 под контролем бета-2-тубулинового промотора, за которой следует 3’ UTR Cop1:TCTTTCTTGCGCTATGACACTTCCAGCAAAAGGTAGGGCGGGCTGCGAGACGGCTTCCCGGCGCTGCATGCAACACCGATGATGCTTCGACCCCCCGAAGCTCCTTCGGGGCTGCATGGGCGCTCCGATGCCGCTCCAGGGCGAGCGCTGTTTAAATAGCCAGGCCCCCGATTGCAAAGACATTATAGCGAGCTACCAAAGCCATATTCAAACACCTAGATCACTACCACTTCTACACAGGCCACTCGAGCTTGTGATCGCACTCCGCTAAGGGGGCGCCTCTTCCTCTTCGTTTCAGTCACAACCCGCAAACATGTACCCATACGATGTTCCAGATTACGCTTCGCCGAAGAAAAAGCGCAAGGTCGAAGCGTCCGACAAGAAGTACAGCATCGGCCTGGACATCGGCACCAACTCTGTGGGCTGGGCCGTGATCACCGACGAGTACAAGGTGCCCAGCAAGAAATTCAAGGTGCTGGGCAACACCGACCGGCACAGCATCAAGAAGAACCTGATCGGAGCCCTGCTGTTCGACAGCGGCGAAACAGCCGAGGCCACCCGGCTGAAGAGAACCGCCAGAAGAAGATACACCAGACGGAAGAACCGGATCTGCTATCTGCAAGAGATCTTCAGCAACGAGATGGCCAAGGTGGACGACAGCTTCTTCCACAGACTGGAAGAGTCCTTCCTGGTGGAAGAGGATAAGAAGCACGAGCGGCACCCCATCTTCGGCAACATCGTGGACGAGGTGGCCTACCACGAGAAGTACCCCACCATCTACCACCTGAGAAAGAAACTGGTGGACAGCACCGACAAGGCCGACCTGCGGCTGATCTATCTGGCCCTGGCCCACATGATCAAGTTCCGGGGCCACTTCCTGATCGAGGGCGACCTGAACCCCGACAACAGCGACGTGGACAAGCTGTTCATCCAGCTGGTGCAGACCTACAACCAGCTGTTCGAGGAAAACCCCATCAACGCCAGCGGCGTGGACGCCAAGGCCATCCTGTCTGCCAGACTGAGCAAGAGCAGACGGCTGGAAAATCTGATCGCCCAGCTGCCCGGCGAGAAGAAGAATGGCCTGTTCGGCAACCTGATTGCCCTGAGCCTGGGCCTGACCCCCAACTTCAAGAGCAACTTCGACCTGGCCGAGGATGCCAAACTGCAGCTGAGCAAGGACACCTACGACGACGACCTGGACAACCTGCTGGCCCAGATCGGCGACCAGTACGCCGACCTGTTTCTGGCCGCCAAGAACCTGTCCGACGCCATCCTGCTGAGCGACATCCTGAGAGTGAACACCGAGATCACCAAGGCCCCCCTGAGCGCCTCTATGATCAAGAGATACGACGAGCACCACCAGGACCTGACCCTGCTGAAAGCTCTCGTGCGGCAGCAGCTGCCTGAGAAGTACAAAGAGATTTTCTTCGACCAGAGCAAGAACGGCTACGCCGGCTACATTGACGGCGGAGCCAGCCAGGAAGAGTTCTACAAGTTCATCAAGCCCATCCTGGAAAAGATGGACGGCACCGAGGAACTGCTCGTGAAGCTGAACAGAGAGGACCTGCTGCGGAAGCAGCGGACCTTCGACAACGGCAGCATCCCCCACCAGATCCACCTGGGAGAGCTGCACGCCATTCTGCGGCGGCAGGAAGATTTTTACCCATTCCTGAAGGACAACCGGGAAAAGATCGAGAAGATCCTGACCTTCCGCATCCCCTACTACGTGGGCCCTCTGGCCAGGGGAAACAGCAGATTCGCCTGGATGACCAGAAAGAGCGAGGAAACCATCACCCCCTGGAACTTCGAGGAAGTGGTGGACAAGGGCGCTTCCGCCCAGAGCTTCATCGAGCGGATGACCAACTTCGATAAGAACCTGCCCAACGAGAAGGTGCTGCCCAAGCACAGCCTGCTGTACGAGTACTTCACCGTGTATAACGAGCTGACCAAAGTGAAATACGTGACCGAGGGAATGAGAAAGCCCGCCTTCCTGAGCGGCGAGCAGAAAAAGGCCATCGTGGACCTGCTGTTCAAGACCAACCGGAAAGTGACCGTGAAGCAGCTGAAAGAGGACTACTTCAAGAAAATCGAGTGCTTCGACTCCGTGGAAATCTCCGGCGTGGAAGATCGGTTCAACGCCTCCCTGGGCACATACCACGATCTGCTGAAAATTATCAAGGACAAGGACTTCCTGGACAATGAGGAAAACGAGGACATTCTGGAAGATATCGTGCTGACCCTGACACTGTTTGAGGACAGAGAGATGATCGAGGAACGGCTGAAAACCTATGCCCACCTGTTCGACGACAAAGTGATGAAGCAGCTGAAGCGGCGGAGATACACCGGCTGGGGCAGGCTGAGCCGGAAGCTGATCAACGGCATCCGGGACAAGCAGTCCGGCAAGACAATCCTGGATTTCCTGAAGTCCGACGGCTTCGCCAACAGAAACTTCATGCAGCTGATCCACGACGACAGCCTGACCTTTAAAGAGGACATCCAGAAAGCCCAGGTGTCCGGCCAGGGCGATAGCCTGCACGAGCACATTGCCAATCTGGCCGGCAGCCCCGCCATTAAGAAGGGCATCCTGCAGACAGTGAAGGTGGTGGACGAGCTCGTGAAAGTGATGGGCCGGCACAAGCCCGAGAACATCGTGATCGAAATGGCCAGAGAGAACCAGACCACCCAGAAGGGACAGAAGAACAGCCGCGAGAGAATGAAGCGGATCGAAGAGGGCATCAAAGAGCTGGGCAGCCAGATCCTGAAAGAACACCCCGTGGAAAACACCCAGCTGCAGAACGAGAAGCTGTACCTGTACTACCTGCAGAATGGGCGGGATATGTACGTGGACCAGGAACTGGACATCAACCGGCTGTCCGACTACGATGTGGACCATATCGTGCCTCAGAGCTTTCTGAAGGACGACTCCATCGACAACAAGGTGCTGACCAGAAGCGACAAGAACCGGGGCAAGAGCGACAACGTGCCCTCCGAAGAGGTCGTGAAGAAGATGAAGAACTACTGGCGGCAGCTGCTGAACGCCAAGCTGATTACCCAGAGAAAGTTCGACAATCTGACCAAGGCCGAGAGAGGCGGCCTGAGCGAACTGGATAAGGCCGGCTTCATCAAGAGACAGCTGGTGGAAACCCGGCAGATCACAAAGCACGTGGCACAGATCCTGGACTCCCGGATGAACACTAAGTACGACGAGAATGACAAGCTGATCCGGGAAGTGAAAGTGATCACCCTGAAGTCCAAGCTGGTGTCCGATTTCCGGAAGGATTTCCAGTTTTACAAAGTGCGCGAGATCAACAACTACCACCACGCCCACGACGCCTACCTGAACGCCGTCGTGGGAACCGCCCTGATCAAAAAGTACCCTAAGCTGGAAAGCGAGTTCGTGTACGGCGACTACAAGGTGTACGACGTGCGGAAGATGATCGCCAAGAGCGAGCAGGAAATCGGCAAGGCTACCGCCAAGTACTTCTTCTACAGCAACATCATGAACTTTTTCAAGACCGAGATTACCCTGGCCAACGGCGAGATCCGGAAGCGGCCTCTGATCGAGACAAACGGCGAAACCGGGGAGATCGTGTGGGATAAGGGCCGGGATTTTGCCACCGTGCGGAAAGTGCTGAGCATGCCCCAAGTGAATATCGTGAAAAAGACCGAGGTGCAGACAGGCGGCTTCAGCAAAGAGTCTATCCTGCCCAAGAGGAACAGCGATAAGCTGATCGCCAGAAAGAAGGACTGGGACCCTAAGAAGTACGGCGGCTTCGACAGCCCCACCGTGGCCTATTCTGTGCTGGTGGTGGCCAAAGTGGAAAAGGGCAAGTCCAAGAAACTGAAGAGTGTGAAAGAGCTGCTGGGGATCACCATCATGGAAAGAAGCAGCTTCGAGAAGAATCCCATCGACTTTCTGGAAGCCAAGGGCTACAAAGAAGTGAAAAAGGACCTGATCATCAAGCTGCCTAAGTACTCCCTGTTCGAGCTGGAAAACGGCCGGAAGAGAATGCTGGCCTCTGCCGGCGAACTGCAGAAGGGAAACGAACTGGCCCTGCCCTCCAAATATGTGAACTTCCTGTACCTGGCCAGCCACTATGAGAAGCTGAAGGGCTCCCCCGAGGATAATGAGCAGAAACAGCTGTTTGTGGAACAGCACAAGCACTACCTGGACGAGATCATCGAGCAGATCAGCGAGTTCTCCAAGAGAGTGATCCTGGCCGACGCTAATCTGGACAAAGTGCTGTCCGCCTACAACAAGCACCGGGATAAGCCCATCAGAGAGCAGGCCGAGAATATCATCCACCTGTTTACCCTGACCAATCTGGGAGCCCCTGCCGCCTTCAAGTACTTTGACACCACCATCGACCGGAAGAGGTACACCAGCACCAAAGAGGTGCTGGACGCCACCCTGATCCACCAGAGCATCACCGGCCTGTACGAGACACGGATCGACCTGTCTCAGCTGGGAGGCGACAGCCCCAAGAAGAAGAGAAAGGTGGAGGCCAGCTAAGGATCCGGCAAGACTGGCCCCGCTTGGCAACGCAACAGTGAGCCCCTCCCTAGTGTGTTTGGGGATGTGACTATGTATTCGTGTGTTGGCCAACGGGTCAACCCGAACAGATTGATACCCGCCTTGGCATTTCCTGTCAGAATGTAACGTCAGTTGATGGTACT (SEQ ID NO: 19).
Последовательность для кассеты, управляющей экспрессией полимеразы T7 под контролем бета-2-тубулинового промотора, за которой следует 3’ UTR Cop1:
TCTTTCTTGCGCTATGACACTTCCAGCAAAAGGTAGGGCGGGCTGCGAGACGGCTTCCCGGCGCTGCATGCAACACCGATGATGCTTCGACCCCCCGAAGCTCCTTCGGGGCTGCATGGGCGCTCCGATGCCGCTCCAGGGCGAGCGCTGTTTAAATAGCCAGGCCCCCGATTGCAAAGACATTATAGCGAGCTACCAAAGCCATATTCAAACACCTAGATCACTACCACTTCTACACAGGCCACTCGAGCTTGTGATCGCACTCCGCTAAGGGGGCGCCTCTTCCTCTTCGTTTCAGTCACAACCCGCAAACatgcctaagaagaagaggaaggttaacacgattaacatcgctaagaacgacttctctgacatcgaactggctgctatcccgttcaacactctggctgaccattacggtgagcgtttagctcgcgaacagttggcccttgagcatgagtcttacgagatgggtgaagcacgcttccgcaagatgtttgagcgtcaacttaaagctggtgaggttgcggataacgctgccgccaagcctctcatcactaccctactccctaagatgattgcacgcatcaacgactggtttgaggaagtgaaagctaagcgcggcaagcgcccgacagccttccagttcctgcaagaaatcaagccggaagccgtagcgtacatcaccattaagaccactctggcttgcctaaccagtgctgacaatacaaccgttcaggctgtagcaagcgcaatcggtcgggccattgaggacgaggctcgcttcggtcgtatccgtgaccttgaagctaagcacttcaagaaaaacgttgaggaacaactcaacaagcgcgtagggcacgtctacaagaaagcatttatgcaagttgtcgaggctgacatgctctctaagggtctactcggtggcgaggcgtggtcttcgtggcataaggaagactctattcatgtaggagtacgctgcatcgagatgctcattgagtcaaccggaatggttagcttacaccgccaaaatgctggcgtagtaggtcaagactctgagactatcgaactcgcacctgaatacgctgaggctatcgcaacccgtgcaggtgcgctggctggcatctctccgatgttccaaccttgcgtagttcctcctaagccgtggactggcattactggtggtggctattgggctaacggtcgtcgtcctctggcgctggtgcgtactcacagtaagaaagcactgatgcgctacgaagacgtttacatgcctgaggtgtacaaagcgattaacattgcgcaaaacaccgcatggaaaatcaacaagaaagtcctagcggtcgccaacgtaatcaccaagtggaagcattgtccggtcgaggacatccctgcgattgagcgtgaagaactcccgatgaaaccggaagacatcgacatgaatcctgaggctctcaccgcgtggaaacgtgctgccgctgctgtgtaccgcaaggacaaggctcgcaagtctcgccgtatcagccttgagttcatgcttgagcaagccaataagtttgctaaccataaggccatctggttcccttacaacatggactggcgcggtcgtgtttacgctgtgtcaatgttcaacccgcaaggtaacgatatgaccaaaggactgcttacgctggcgaaaggtaaaccaatcggtaaggaaggttactactggctgaaaatccacggtgcaaactgtgcgggtgtcgacaaggttccgttccctgagcgcatcaagttcattgaggaaaaccacgagaacatcatggcttgcgctaagtctccactggagaacacttggtgggctgagcaagattctccgttctgcttccttgcgttctgctttgagtacgctggggtacagcaccacggcctgagctataactgctcccttccgctggcgtttgacgggtcttgctctggcatccagcacttctccgcgatgctccgagatgaggtaggtggtcgcgcggttaacttgcttcctagtgaaaccgttcaggacatctacgggattgttgctaagaaagtcaacgagattctacaagcagacgcaatcaatgggaccgataacgaagtagttaccgtgaccgatgagaacactggtgaaatctctgagaaagtcaagctgggcactaaggcactggctggtcaatggctggcttacggtgttactcgcagtgtgactaagcgttcagtcatgacgctggcttacgggtccaaagagttcggcttccgtcaacaagtgctggaagataccattcagccagctattgattccggcaagggtctgatgttcactcagccgaatcaggctgctggatacatggctaagctgatttgggaatctgtgagcgtgacggtggtagctgcggttgaagcaatgaactggcttaagtctgctgctaagctgctggctgctgaggtcaaagataagaagactggagagattcttcgcaagcgttgcgctgtgcattgggtaactcctgatggtttccctgtgtggcaggaatacaagaagcctattcagacgcgcttgaacctgatgttcctcggtcagttccgcttacagcctaccattaacaccaacaaagatagcgagattgatgcacacaaacaggagtctggtatcgctcctaactttgtacacagccaagacggtagccaccttcgtaagactgtagtgtgggcacacgagaagtacggaatcgaatcttttgcactgattcacgactccttcggtacgattccggctgacgctgcgaacctgttcaaagcagtgcgcgaaactatggttgacacatatgagtcttgtgatgtactggctgatttctacgaccagttcgctgaccagttgcacgagtctcaattggacaaaatgccagcacttccggctaaaggtaacttgaacctccgtgacatcttagagtcggacttcgcgttcgcgtaaGGATCCGGCAAGACTGGCCCCGCTTGGCAACGCAACAGTGAGCCCCTCCCTAGTGTGTTTGGGGATGTGACTATGTATTCGTGTGTTGGCCAACGGGTCAACCCGAACAGATTGATACCCGCCTTGGCATTTCCTGTCAGAATGTAACGTCAGTTGATGGTACT (SEQ ID NO: 20).
Последовательность направляющей РНК, управляемая промотором T7 (промотор T7, N представляют собой нацеливающую последовательность):
gaaatTAATACGACTCACTATANNNNNNNNNNNNNNNNNNNNgttttagagctaGAAAtagcaagttaaaataaggctagtccgttatcaacttgaaaaagtggcaccgagtcggtgcttttttt (SEQ ID NO: 21).
Доставке гена
Штаммы CC-124 и CC-125 Chlamydomonas reinhardtii из Ресурсного центра Chlamydomonas (Chlamydomonas Resource Center) будут использоваться для электропорации. Протокол электропорации соответствует стандартному рекомендованному протоколу из набора GeneArt Chlamydomonas Engineering kit.
Заявители также получали линию Chlamydomonas reinhardtii, которые экспрессируют Cas9 конститутивно. Это можно выполнить при помощи pChlamy1 (линеаризованная с использованием PvuI) и отбора в отношении устойчивых к гигромицину колоний. Последовательность pChlamy1, содержащая Cas9, представлена ниже. Таким образом, для достижения нокаутирования гена необходимо просто доставить РНК для направляющей РНК. Для гомологичной рекомбинации заявители доставляли направляющую РНК, а также линеаризованную матрицу для гомологичной рекомбинации.
pChlamy1-Cas9:
TGCGGTATTTCACACCGCATCAGGTGGCACTTTTCGGGGAAATGTGCGCGGAACCCCTATTTGTTTATTTTTCTAAATACATTCAAATATGTATCCGCTCATGAGATTATCAAAAAGGATCTTCACCTAGATCCTTTTAAATTAAAAATGAAGTTTTAAATCAATCTAAAGTATATATGAGTAAACTTGGTCTGACAGTTACCAATGCTTAATCAGTGAGGCACCTATCTCAGCGATCTGTCTATTTCGTTCATCCATAGTTGCCTGACTCCCCGTCGTGTAGATAACTACGATACGGGAGGGCTTACCATCTGGCCCCAGTGCTGCAATGATACCGCGAGACCCACGCTCACCGGCTCCAGATTTATCAGCAATAAACCAGCCAGCCGGAAGGGCCGAGCGCAGAAGTGGTCCTGCAACTTTATCCGCCTCCATCCAGTCTATTAATTGTTGCCGGGAAGCTAGAGTAAGTAGTTCGCCAGTTAATAGTTTGCGCAACGTTGTTGCCATTGCTACAGGCATCGTGGTGTCACGCTCGTCGTTTGGTATGGCTTCATTCAGCTCCGGTTCCCAACGATCAAGGCGAGTTACATGATCCCCCATGTTGTGCAAAAAAGCGGTTAGCTCCTTCGGTCCTCCGATCGTTGTCAGAAGTAAGTTGGCCGCAGTGTTATCACTCATGGTTATGGCAGCACTGCATAATTCTCTTACTGTCATGCCATCCGTAAGATGCTTTTCTGTGACTGGTGAGTACTCAACCAAGTCATTCTGAGAATAGTGTATGCGGCGACCGAGTTGCTCTTGCCCGGCGTCAATACGGGATAATACCGCGCCACATAGCAGAACTTTAAAAGTGCTCATCATTGGAAAACGTTCTTCGGGGCGAAAACTCTCAAGGATCTTACCGCTGTTGAGATCCAGTTCGATGTAACCCACTCGTGCACCCAACTGATCTTCAGCATCTTTTACTTTCACCAGCGTTTCTGGGTGAGCAAAAACAGGAAGGCAAAATGCCGCAAAAAAGGGAATAAGGGCGACACGGAAATGTTGAATACTCATACTCTTCCTTTTTCAATATTATTGAAGCATTTATCAGGGTTATTGTCTCATGACCAAAATCCCTTAACGTGAGTTTTCGTTCCACTGAGCGTCAGACCCCGTAGAAAAGATCAAAGGATCTTCTTGAGATCCTTTTTTTCTGCGCGTAATCTGCTGCTTGCAAACAAAAAAACCACCGCTACCAGCGGTGGTTTGTTTGCCGGATCAAGAGCTACCAACTCTTTTTCCGAAGGTAACTGGCTTCAGCAGAGCGCAGATACCAAATACTGTTCTTCTAGTGTAGCCGTAGTTAGGCCACCACTTCAAGAACTCTGTAGCACCGCCTACATACCTCGCTCTGCTAATCCTGTTACCAGTGGCTGTTGCCAGTGGCGATAAGTCGTGTCTTACCGGGTTGGACTCAAGACGATAGTTACCGGATAAGGCGCAGCGGTCGGGCTGAACGGGGGGTTCGTGCACACAGCCCAGCTTGGAGCGAACGACCTACACCGAACTGAGATACCTACAGCGTGAGCTATGAGAAAGCGCCACGCTTCCCGAAGGGAGAAAGGCGGACAGGTATCCGGTAAGCGGCAGGGTCGGAACAGGAGAGCGCACGAGGGAGCTTCCAGGGGGAAACGCCTGGTATCTTTATAGTCCTGTCGGGTTTCGCCACCTCTGACTTGAGCGTCGATTTTTGTGATGCTCGTCAGGGGGGCGGAGCCTATGGAAAAACGCCAGCAACGCGGCCTTTTTACGGTTCCTGGCCTTTTGCTGGCCTTTTGCTCACATGTTCTTTCCTGCGTTATCCCCTGATTCTGTGGATAACCGTATTACCGCCTTTGAGTGAGCTGATACCGCTCGCCGCAGCCGAACGACCGAGCGCAGCGAGTCAGTGAGCGAGGAAGCGGTCGCTGAGGCTTGACATGATTGGTGCGTATGTTTGTATGAAGCTACAGGACTGATTTGGCGGGCTATGAGGGCGGGGGAAGCTCTGGAAGGGCCGCGATGGGGCGCGCGGCGTCCAGAAGGCGCCATACGGCCCGCTGGCGGCACCCATCCGGTATAAAAGCCCGCGACCCCGAACGGTGACCTCCACTTTCAGCGACAAACGAGCACTTATACATACGCGACTATTCTGCCGCTATACATAACCACTCAGCTAGCTTAAGATCCCATCAAGCTTGCATGCCGGGCGCGCCAGAAGGAGCGCAGCCAAACCAGGATGATGTTTGATGGGGTATTTGAGCACTTGCAACCCTTATCCGGAAGCCCCCTGGCCCACAAAGGCTAGGCGCCAATGCAAGCAGTTCGCATGCAGCCCCTGGAGCGGTGCCCTCCTGATAAACCGGCCAGGGGGCCTATGTTCTTTACTTTTTTACAAGAGAAGTCACTCAACATCTTAAAATGGCCAGGTGAGTCGACGAGCAAGCCCGGCGGATCAGGCAGCGTGCTTGCAGATTTGACTTGCAACGCCCGCATTGTGTCGACGAAGGCTTTTGGCTCCTCTGTCGCTGTCTCAAGCAGCATCTAACCCTGCGTCGCCGTTTCCATTTGCAGGAGATTCGAGGTACCATGTACCCATACGATGTTCCAGATTACGCTTCGCCGAAGAAAAAGCGCAAGGTCGAAGCGTCCGACAAGAAGTACAGCATCGGCCTGGACATCGGCACCAACTCTGTGGGCTGGGCCGTGATCACCGACGAGTACAAGGTGCCCAGCAAGAAATTCAAGGTGCTGGGCAACACCGACCGGCACAGCATCAAGAAGAACCTGATCGGAGCCCTGCTGTTCGACAGCGGCGAAACAGCCGAGGCCACCCGGCTGAAGAGAACCGCCAGAAGAAGATACACCAGACGGAAGAACCGGATCTGCTATCTGCAAGAGATCTTCAGCAACGAGATGGCCAAGGTGGACGACAGCTTCTTCCACAGACTGGAAGAGTCCTTCCTGGTGGAAGAGGATAAGAAGCACGAGCGGCACCCCATCTTCGGCAACATCGTGGACGAGGTGGCCTACCACGAGAAGTACCCCACCATCTACCACCTGAGAAAGAAACTGGTGGACAGCACCGACAAGGCCGACCTGCGGCTGATCTATCTGGCCCTGGCCCACATGATCAAGTTCCGGGGCCACTTCCTGATCGAGGGCGACCTGAACCCCGACAACAGCGACGTGGACAAGCTGTTCATCCAGCTGGTGCAGACCTACAACCAGCTGTTCGAGGAAAACCCCATCAACGCCAGCGGCGTGGACGCCAAGGCCATCCTGTCTGCCAGACTGAGCAAGAGCAGACGGCTGGAAAATCTGATCGCCCAGCTGCCCGGCGAGAAGAAGAATGGCCTGTTCGGCAACCTGATTGCCCTGAGCCTGGGCCTGACCCCCAACTTCAAGAGCAACTTCGACCTGGCCGAGGATGCCAAACTGCAGCTGAGCAAGGACACCTACGACGACGACCTGGACAACCTGCTGGCCCAGATCGGCGACCAGTACGCCGACCTGTTTCTGGCCGCCAAGAACCTGTCCGACGCCATCCTGCTGAGCGACATCCTGAGAGTGAACACCGAGATCACCAAGGCCCCCCTGAGCGCCTCTATGATCAAGAGATACGACGAGCACCACCAGGACCTGACCCTGCTGAAAGCTCTCGTGCGGCAGCAGCTGCCTGAGAAGTACAAAGAGATTTTCTTCGACCAGAGCAAGAACGGCTACGCCGGCTACATTGACGGCGGAGCCAGCCAGGAAGAGTTCTACAAGTTCATCAAGCCCATCCTGGAAAAGATGGACGGCACCGAGGAACTGCTCGTGAAGCTGAACAGAGAGGACCTGCTGCGGAAGCAGCGGACCTTCGACAACGGCAGCATCCCCCACCAGATCCACCTGGGAGAGCTGCACGCCATTCTGCGGCGGCAGGAAGATTTTTACCCATTCCTGAAGGACAACCGGGAAAAGATCGAGAAGATCCTGACCTTCCGCATCCCCTACTACGTGGGCCCTCTGGCCAGGGGAAACAGCAGATTCGCCTGGATGACCAGAAAGAGCGAGGAAACCATCACCCCCTGGAACTTCGAGGAAGTGGTGGACAAGGGCGCTTCCGCCCAGAGCTTCATCGAGCGGATGACCAACTTCGATAAGAACCTGCCCAACGAGAAGGTGCTGCCCAAGCACAGCCTGCTGTACGAGTACTTCACCGTGTATAACGAGCTGACCAAAGTGAAATACGTGACCGAGGGAATGAGAAAGCCCGCCTTCCTGAGCGGCGAGCAGAAAAAGGCCATCGTGGACCTGCTGTTCAAGACCAACCGGAAAGTGACCGTGAAGCAGCTGAAAGAGGACTACTTCAAGAAAATCGAGTGCTTCGACTCCGTGGAAATCTCCGGCGTGGAAGATCGGTTCAACGCCTCCCTGGGCACATACCACGATCTGCTGAAAATTATCAAGGACAAGGACTTCCTGGACAATGAGGAAAACGAGGACATTCTGGAAGATATCGTGCTGACCCTGACACTGTTTGAGGACAGAGAGATGATCGAGGAACGGCTGAAAACCTATGCCCACCTGTTCGACGACAAAGTGATGAAGCAGCTGAAGCGGCGGAGATACACCGGCTGGGGCAGGCTGAGCCGGAAGCTGATCAACGGCATCCGGGACAAGCAGTCCGGCAAGACAATCCTGGATTTCCTGAAGTCCGACGGCTTCGCCAACAGAAACTTCATGCAGCTGATCCACGACGACAGCCTGACCTTTAAAGAGGACATCCAGAAAGCCCAGGTGTCCGGCCAGGGCGATAGCCTGCACGAGCACATTGCCAATCTGGCCGGCAGCCCCGCCATTAAGAAGGGCATCCTGCAGACAGTGAAGGTGGTGGACGAGCTCGTGAAAGTGATGGGCCGGCACAAGCCCGAGAACATCGTGATCGAAATGGCCAGAGAGAACCAGACCACCCAGAAGGGACAGAAGAACAGCCGCGAGAGAATGAAGCGGATCGAAGAGGGCATCAAAGAGCTGGGCAGCCAGATCCTGAAAGAACACCCCGTGGAAAACACCCAGCTGCAGAACGAGAAGCTGTACCTGTACTACCTGCAGAATGGGCGGGATATGTACGTGGACCAGGAACTGGACATCAACCGGCTGTCCGACTACGATGTGGACCATATCGTGCCTCAGAGCTTTCTGAAGGACGACTCCATCGACAACAAGGTGCTGACCAGAAGCGACAAGAACCGGGGCAAGAGCGACAACGTGCCCTCCGAAGAGGTCGTGAAGAAGATGAAGAACTACTGGCGGCAGCTGCTGAACGCCAAGCTGATTACCCAGAGAAAGTTCGACAATCTGACCAAGGCCGAGAGAGGCGGCCTGAGCGAACTGGATAAGGCCGGCTTCATCAAGAGACAGCTGGTGGAAACCCGGCAGATCACAAAGCACGTGGCACAGATCCTGGACTCCCGGATGAACACTAAGTACGACGAGAATGACAAGCTGATCCGGGAAGTGAAAGTGATCACCCTGAAGTCCAAGCTGGTGTCCGATTTCCGGAAGGATTTCCAGTTTTACAAAGTGCGCGAGATCAACAACTACCACCACGCCCACGACGCCTACCTGAACGCCGTCGTGGGAACCGCCCTGATCAAAAAGTACCCTAAGCTGGAAAGCGAGTTCGTGTACGGCGACTACAAGGTGTACGACGTGCGGAAGATGATCGCCAAGAGCGAGCAGGAAATCGGCAAGGCTACCGCCAAGTACTTCTTCTACAGCAACATCATGAACTTTTTCAAGACCGAGATTACCCTGGCCAACGGCGAGATCCGGAAGCGGCCTCTGATCGAGACAAACGGCGAAACCGGGGAGATCGTGTGGGATAAGGGCCGGGATTTTGCCACCGTGCGGAAAGTGCTGAGCATGCCCCAAGTGAATATCGTGAAAAAGACCGAGGTGCAGACAGGCGGCTTCAGCAAAGAGTCTATCCTGCCCAAGAGGAACAGCGATAAGCTGATCGCCAGAAAGAAGGACTGGGACCCTAAGAAGTACGGCGGCTTCGACAGCCCCACCGTGGCCTATTCTGTGCTGGTGGTGGCCAAAGTGGAAAAGGGCAAGTCCAAGAAACTGAAGAGTGTGAAAGAGCTGCTGGGGATCACCATCATGGAAAGAAGCAGCTTCGAGAAGAATCCCATCGACTTTCTGGAAGCCAAGGGCTACAAAGAAGTGAAAAAGGACCTGATCATCAAGCTGCCTAAGTACTCCCTGTTCGAGCTGGAAAACGGCCGGAAGAGAATGCTGGCCTCTGCCGGCGAACTGCAGAAGGGAAACGAACTGGCCCTGCCCTCCAAATATGTGAACTTCCTGTACCTGGCCAGCCACTATGAGAAGCTGAAGGGCTCCCCCGAGGATAATGAGCAGAAACAGCTGTTTGTGGAACAGCACAAGCACTACCTGGACGAGATCATCGAGCAGATCAGCGAGTTCTCCAAGAGAGTGATCCTGGCCGACGCTAATCTGGACAAAGTGCTGTCCGCCTACAACAAGCACCGGGATAAGCCCATCAGAGAGCAGGCCGAGAATATCATCCACCTGTTTACCCTGACCAATCTGGGAGCCCCTGCCGCCTTCAAGTACTTTGACACCACCATCGACCGGAAGAGGTACACCAGCACCAAAGAGGTGCTGGACGCCACCCTGATCCACCAGAGCATCACCGGCCTGTACGAGACACGGATCGACCTGTCTCAGCTGGGAGGCGACAGCCCCAAGAAGAAGAGAAAGGTGGAGGCCAGCTAACATATGATTCGAATGTCTTTCTTGCGCTATGACACTTCCAGCAAAAGGTAGGGCGGGCTGCGAGACGGCTTCCCGGCGCTGCATGCAACACCGATGATGCTTCGACCCCCCGAAGCTCCTTCGGGGCTGCATGGGCGCTCCGATGCCGCTCCAGGGCGAGCGCTGTTTAAATAGCCAGGCCCCCGATTGCAAAGACATTATAGCGAGCTACCAAAGCCATATTCAAACACCTAGATCACTACCACTTCTACACAGGCCACTCGAGCTTGTGATCGCACTCCGCTAAGGGGGCGCCTCTTCCTCTTCGTTTCAGTCACAACCCGCAAACATGACACAAGAATCCCTGTTACTTCTCGACCGTATTGATTCGGATGATTCCTACGCGAGCCTGCGGAACGACCAGGAATTCTGGGAGGTGAGTCGACGAGCAAGCCCGGCGGATCAGGCAGCGTGCTTGCAGATTTGACTTGCAACGCCCGCATTGTGTCGACGAAGGCTTTTGGCTCCTCTGTCGCTGTCTCAAGCAGCATCTAACCCTGCGTCGCCGTTTCCATTTGCAGCCGCTGGCCCGCCGAGCCCTGGAGGAGCTCGGGCTGCCGGTGCCGCCGGTGCTGCGGGTGCCCGGCGAGAGCACCAACCCCGTACTGGTCGGCGAGCCCGGCCCGGTGATCAAGCTGTTCGGCGAGCACTGGTGCGGTCCGGAGAGCCTCGCGTCGGAGTCGGAGGCGTACGCGGTCCTGGCGGACGCCCCGGTGCCGGTGCCCCGCCTCCTCGGCCGCGGCGAGCTGCGGCCCGGCACCGGAGCCTGGCCGTGGCCCTACCTGGTGATGAGCCGGATGACCGGCACCACCTGGCGGTCCGCGATGGACGGCACGACCGACCGGAACGCGCTGCTCGCCCTGGCCCGCGAACTCGGCCGGGTGCTCGGCCGGCTGCACAGGGTGCCGCTGACCGGGAACACCGTGCTCACCCCCCATTCCGAGGTCTTCCCGGAACTGCTGCGGGAACGCCGCGCGGCGACCGTCGAGGACCACCGCGGGTGGGGCTACCTCTCGCCCCGGCTGCTGGACCGCCTGGAGGACTGGCTGCCGGACGTGGACACGCTGCTGGCCGGCCGCGAACCCCGGTTCGTCCACGGCGACCTGCACGGGACCAACATCTTCGTGGACCTGGCCGCGACCGAGGTCACCGGGATCGTCGACTTCACCGACGTCTATGCGGGAGACTCCCGCTACAGCCTGGTGCAACTGCATCTCAACGCCTTCCGGGGCGACCGCGAGATCCTGGCCGCGCTGCTCGACGGGGCGCAGTGGAAGCGGACCGAGGACTTCGCCCGCGAACTGCTCGCCTTCACCTTCCTGCACGACTTCGAGGTGTTCGAGGAGACCCCGCTGGATCTCTCCGGCTTCACCGATCCGGAGGAACTGGCGCAGTTCCTCTGGGGGCCGCCGGACACCGCCCCCGGCGCCTGATAAGGATCCGGCAAGACTGGCCCCGCTTGGCAACGCAACAGTGAGCCCCTCCCTAGTGTGTTTGGGGATGTGACTATGTATTCGTGTGTTGGCCAACGGGTCAACCCGAACAGATTGATACCCGCCTTGGCATTTCCTGTCAGAATGTAACGTCAGTTGATGGTACT (SEQ ID NO: 22).
Для всех модифицированных клеток Chlamydomonas reinhardtii заявители использовали ПЦР, анализ с помощью нуклеазы SURVEYOR и секвенирование ДНК для подтверждения успешной модификации.
Несмотря на то, что предпочтительные варианты осуществления настоящего изобретения были показаны и описаны в данном документе, для специалиста в данной области будет очевидно, что такие варианты осуществления предоставлены только в качестве примера. Многочисленные вариации, изменения и замены теперь будут очевидны для специалиста в данной области без отступления от сути настоящего изобретения. Следует понимать, что различные альтернативные варианты вариантов осуществления настоящего изобретения, раскрытые в данном документе, можно применять при практическом осуществлении настоящего изобретения. Предполагают, что следующая формула изобретения определяет объем настоящего изобретения, и что, таким образом, охвачены способы и структуры в пределах объема данной формулы изобретения и их эквиваленты.
Библиографические ссылки:
1. Urnov, F.D., Rebar, E.J., Holmes, M.C., Zhang, H.S. & Gregory, P.D. Genome editing with engineered zinc finger nucleases. Nat. Rev. Genet. 11, 636-646 (2010).
2. Bogdanove, A.J. & Voytas, D.F. TAL effectors: customizable proteins for DNA targeting. Science 333, 1843-1846 (2011).
3. Stoddard, B.L. Homing endonuclease structure and function. Q. Rev. Biophys. 38, 49-95 (2005).
4. Bae, T. & Schneewind, O. Allelic replacement in Staphylococcus aureus with inducible counter-selection. Plasmid 55, 58-63 (2006).
5. Sung, C.K., Li, H., Claverys, J.P. & Morrison, D.A. An rpsL cassette, janus, for gene replacement through negative selection in Streptococcus pneumoniae. Appl. Environ. Microbiol. 67, 5190-5196 (2001).
6. Sharan, S.K., Thomason, L.C., Kuznetsov, S.G. & Court, D.L. Recombineering: a homologous recombination-based method of genetic engineering. Nat. Protoc. 4, 206-223 (2009).
7. Jinek, M. et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science 337, 816-821 (2012).
8. Deveau, H., Garneau, J.E. & Moineau, S. CRISPR/Cas system and its role in phage-bacteria interactions. Annu. Rev. Microbiol. 64, 475-493 (2010).
9. Horvath, P. & Barrangou, R. CRISPR/Cas, the immune system of bacteria and archaea. Science 327, 167-170 (2010).
10. Terns, M.P. & Terns, R.M. CRISPR-based adaptive immune systems. Curr. Opin. Microbiol. 14, 321-327 (2011).
11. van der Oost, J., Jore, M.M., Westra, E.R., Lundgren, M. & Brouns, S.J. CRISPR-based adaptive and heritable immunity in prokaryotes. Trends. Biochem. Sci. 34, 401-407 (2009).
12. Brouns, S.J. et al. Small CRISPR RNAs guide antiviral defense in prokaryotes. Science 321, 960-964 (2008).
13. Carte, J., Wang, R., Li, H., Terns, R.M. & Terns, M.P. Cas6 is an endoribonuclease that generates guide RNAs for invader defense in prokaryotes. Genes Dev. 22, 3489-3496 (2008).
14. Deltcheva, E. et al. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III. Nature 471, 602-607 (2011).
15. Hatoum-Aslan, A., Maniv, I. & Marraffini, L.A. Mature clustered, regularly interspaced, short palindromic repeats RNA (crRNA) length is measured by a ruler mechanism anchored at the precursor processing site. Proc. Natl. Acad. Sci. U.S.A. 108, 21218-21222 (2011).
16. Haurwitz, R.E., Jinek, M., Wiedenheft, B., Zhou, K. & Doudna, J.A. Sequence- and structure-specific RNA processing by a CRISPR endonuclease. Science 329, 1355-1358 (2010).
17. Deveau, H. et al. Phage response to CRISPR-encoded resistance in Streptococcus thermophilus. J. Bacteriol. 190, 1390-1400 (2008).
18. Gasiunas, G., Barrangou, R., Horvath, P. & Siksnys, V. Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria. Proc. Natl. Acad. Sci. U.S.A. (2012).
19. Makarova, K.S., Aravind, L., Wolf, Y.I. & Koonin, E.V. Unification of Cas protein families and a simple scenario for the origin and evolution of CRISPR-Cas systems. Biol. Direct. 6, 38 (2011).
20. Barrangou, R. RNA-mediated programmable DNA cleavage. Nat. Biotechnol. 30, 836-838 (2012).
21. Brouns, S.J. Molecular biology. A Swiss army knife of immunity. Science 337, 808-809 (2012).
22. Carroll, D. A CRISPR Approach to Gene Targeting. Mol. Ther. 20, 1658-1660 (2012).
23. Bikard, D., Hatoum-Aslan, A., Mucida, D. & Marraffini, L.A. CRISPR interference can prevent natural transformation and virulence acquisition during in vivo bacterial infection. Cell Host Microbe 12, 177-186 (2012).
24. Sapranauskas, R. et al. The Streptococcus thermophilus CRISPR/Cas system provides immunity in Escherichia coli. Nucleic Acids Res. (2011).
25. Semenova, E. et al. Interference by clustered regularly interspaced short palindromic repeat (CRISPR) RNA is governed by a seed sequence. Proc. Natl. Acad. Sci. U.S.A. (2011).
26. Wiedenheft, B. et al. RNA-guided complex from a bacterial immune system enhances target recognition through seed sequence interactions. Proc. Natl. Acad. Sci. U.S.A. (2011).
27. Zahner, D. & Hakenbeck, R. The Streptococcus pneumoniae beta-galactosidase is a surface protein. J. Bacteriol. 182, 5919-5921 (2000).
28. Marraffini, L.A., Dedent, A.C. & Schneewind, O. Sortases and the art of anchoring proteins to the envelopes of gram-positive bacteria. Microbiol. Mol. Biol. Rev. 70, 192-221 (2006).
29. Motamedi, M.R., Szigety, S.K. & Rosenberg, S.M. Double-strand-break repair recombination in Escherichia coli: physical evidence for a DNA replication mechanism in vivo. Genes Dev. 13, 2889-2903 (1999).
30. Hosaka, T. et al. The novel mutation K87E in ribosomal protein S12 enhances protein synthesis activity during the late growth phase in Escherichia coli. Mol. Genet. Genomics 271, 317-324 (2004).
31. Costantino, N. & Court, D.L. Enhanced levels of lambda Red-mediated recombinants in mismatch repair mutants. Proc. Natl. Acad. Sci. U.S.A. 100, 15748-15753 (2003).
32. Edgar, R. & Qimron, U. The Escherichia coli CRISPR system protects from lambda lysogenization, lysogens, and prophage induction. J. Bacteriol. 192, 6291-6294 (2010).
33. Marraffini, L.A. & Sontheimer, E.J. Self versus non-self discrimination during CRISPR RNA-directed immunity. Nature 463, 568-571 (2010).
34. Fischer, S. et al. An archaeal immune system can detect multiple Protospacer Adjacent Motifs (PAMs) to target invader DNA. J. Biol. Chem. 287, 33351-33363 (2012).
35. Gudbergsdottir, S. et al. Dynamic properties of the Sulfolobus CRISPR/Cas and CRISPR/Cmr systems when challenged with vector-borne viral and plasmid genes and protospacers. Mol. Microbiol. 79, 35-49 (2011).
36. Wang, H.H. et al. Genome-scale promoter engineering by coselection MAGE. Nat Methods 9, 591-593 (2012).
37. Cong, L. et al. Multiplex Genome Engineering Using CRISPR/Cas Systems. Science в печати (2013).
38. Mali, P. et al. RNA-Guided Human Genome Engineering via Cas9. Science в печати (2013).
39. Hoskins, J. et al. Genome of the bacterium Streptococcus pneumoniae strain R6. J. Bacteriol. 183, 5709-5717 (2001).
40. Havarstein, L.S., Coomaraswamy, G. & Morrison, D.A. An unmodified heptadecapeptide pheromone induces competence for genetic transformation in Streptococcus pneumoniae. Proc. Natl. Acad. Sci. U.S.A. 92, 11140-11144 (1995).
41. Horinouchi, S. & Weisblum, B. Nucleotide sequence and functional map of pC194, a plasmid that specifies inducible chloramphenicol resistance. J. Bacteriol. 150, 815-825 (1982).
42. Horton, R.M. In Vitro Recombination and Mutagenesis of DNA : SOEing Together Tailor-Made Genes. Methods Mol. Biol. 15, 251-261 (1993).
43. Podbielski, A., Spellerberg, B., Woischnik, M., Pohl, B. & Lutticken, R. Novel series of plasmid vectors for gene inactivation and expression analysis in group A streptococci (GAS). Gene 177, 137-147 (1996).
44. Husmann, L.K., Scott, J.R., Lindahl, G. & Stenberg, L. Expression of the Arp protein, a member of the M protein family, is not sufficient to inhibit phagocytosis of Streptococcus pyogenes. Infection and immunity 63, 345-348 (1995).
45. Gibson, D.G. et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nat Methods 6, 343-345 (2009).
46. Garneau J. E. et al. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature 468,67–71(04 November 2010)
47. Barrangou R. et al. CRISPR provides acquired resistance against viruses in prokaryotes. Science. 2007 Mar 23;315(5819):1709-12.
48. Ishino Y. et al. Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product. J Bacteriol. 1987 Dec;169(12):5429-33.
49. Mojica F. J. M et al. Biological significance of a family of regularly spaced repeats in the genomes of Archaea, Bacteria and mitochondria. Molecular Microbiology (2000) 36(1), 244-246.
50. Jansen R. et al. Identification of genes that are associated with DNA repeats in prokaryotes. Molecular Microbiology (2002) 43(6), 1565–1575.
--->
Перечень последовательностей
SEQUENCE LISTING
<110> THE BROAD INSTITUTE, INC.
MASSACHUSETTS INSTITUTE OF TECHNOLOGY
<120> CRISPR-CAS SYSTEMS AND METHODS FOR ALTERING EXPRESSION OF GENE
PRODUCTS
<130> 44790.99.2044
<140> PCT/US2013/074743
<141> 2013-12-12
<150> 14/054,414
<151> 2013-10-15
<150> 61/842,322
<151> 2013-07-02
<150> 61/835,931
<151> 2013-06-17
<150> 61/791,409
<151> 2013-03-15
<150> 61/748,427
<151> 2013-01-02
<150> 61/736,527
<151> 2012-12-12
<160> 181
<170> PatentIn version 3.5
<210> 1
<211> 27
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t or g
<220>
<221> modified_base
<222> (21)..(22)
<223> a, c, t, g, unknown or other
<400> 1
nnnnnnnnnn nnnnnnnnnn nnagaaw 27
<210> 2
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<220>
<221> modified_base
<222> (1)..(12)
<223> a, c, t or g
<220>
<221> modified_base
<222> (13)..(14)
<223> a, c, t, g, unknown or other
<400> 2
nnnnnnnnnn nnnnagaaw 19
<210> 3
<211> 27
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t or g
<220>
<221> modified_base
<222> (21)..(22)
<223> a, c, t, g, unknown or other
<400> 3
nnnnnnnnnn nnnnnnnnnn nnagaaw 27
<210> 4
<211> 18
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<220>
<221> modified_base
<222> (1)..(11)
<223> a, c, t or g
<220>
<221> modified_base
<222> (12)..(13)
<223> a, c, t, g, unknown or other
<400> 4
nnnnnnnnnn nnnagaaw 18
<210> 5
<211> 137
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t, g, unknown or other
<400> 5
nnnnnnnnnn nnnnnnnnnn gtttttgtac tctcaagatt tagaaataaa tcttgcagaa 60
gctacaaaga taaggcttca tgccgaaatc aacaccctgt cattttatgg cagggtgttt 120
tcgttattta atttttt 137
<210> 6
<211> 123
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t, g, unknown or other
<400> 6
nnnnnnnnnn nnnnnnnnnn gtttttgtac tctcagaaat gcagaagcta caaagataag 60
gcttcatgcc gaaatcaaca ccctgtcatt ttatggcagg gtgttttcgt tatttaattt 120
ttt 123
<210> 7
<211> 110
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t, g, unknown or other
<400> 7
nnnnnnnnnn nnnnnnnnnn gtttttgtac tctcagaaat gcagaagcta caaagataag 60
gcttcatgcc gaaatcaaca ccctgtcatt ttatggcagg gtgttttttt 110
<210> 8
<211> 102
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t, g, unknown or other
<400> 8
nnnnnnnnnn nnnnnnnnnn gttttagagc tagaaatagc aagttaaaat aaggctagtc 60
cgttatcaac ttgaaaaagt ggcaccgagt cggtgctttt tt 102
<210> 9
<211> 88
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t, g, unknown or other
<400> 9
nnnnnnnnnn nnnnnnnnnn gttttagagc tagaaatagc aagttaaaat aaggctagtc 60
cgttatcaac ttgaaaaagt gttttttt 88
<210> 10
<211> 76
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t, g, unknown or other
<400> 10
nnnnnnnnnn nnnnnnnnnn gttttagagc tagaaatagc aagttaaaat aaggctagtc 60
cgttatcatt tttttt 76
<210> 11
<211> 12
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 11
gttttagagc ta 12
<210> 12
<211> 31
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 12
tagcaagtta aaataaggct agtccgtttt t 31
<210> 13
<211> 12
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 13
guuuuagagc ua 12
<210> 14
<211> 33
<212> DNA
<213> Homo sapiens
<400> 14
ggacatcgat gtcacctcca atgactaggg tgg 33
<210> 15
<211> 33
<212> DNA
<213> Homo sapiens
<400> 15
cattggaggt gacatcgatg tcctccccat tgg 33
<210> 16
<211> 33
<212> DNA
<213> Homo sapiens
<400> 16
ggaagggcct gagtccgagc agaagaagaa ggg 33
<210> 17
<211> 33
<212> DNA
<213> Homo sapiens
<400> 17
ggtggcgaga ggggccgaga ttgggtgttc agg 33
<210> 18
<211> 33
<212> DNA
<213> Homo sapiens
<400> 18
atgcaggagg gtggcgagag gggccgagat tgg 33
<210> 19
<211> 4677
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 19
tctttcttgc gctatgacac ttccagcaaa aggtagggcg ggctgcgaga cggcttcccg 60
gcgctgcatg caacaccgat gatgcttcga ccccccgaag ctccttcggg gctgcatggg 120
cgctccgatg ccgctccagg gcgagcgctg tttaaatagc caggcccccg attgcaaaga 180
cattatagcg agctaccaaa gccatattca aacacctaga tcactaccac ttctacacag 240
gccactcgag cttgtgatcg cactccgcta agggggcgcc tcttcctctt cgtttcagtc 300
acaacccgca aacatgtacc catacgatgt tccagattac gcttcgccga agaaaaagcg 360
caaggtcgaa gcgtccgaca agaagtacag catcggcctg gacatcggca ccaactctgt 420
gggctgggcc gtgatcaccg acgagtacaa ggtgcccagc aagaaattca aggtgctggg 480
caacaccgac cggcacagca tcaagaagaa cctgatcgga gccctgctgt tcgacagcgg 540
cgaaacagcc gaggccaccc ggctgaagag aaccgccaga agaagataca ccagacggaa 600
gaaccggatc tgctatctgc aagagatctt cagcaacgag atggccaagg tggacgacag 660
cttcttccac agactggaag agtccttcct ggtggaagag gataagaagc acgagcggca 720
ccccatcttc ggcaacatcg tggacgaggt ggcctaccac gagaagtacc ccaccatcta 780
ccacctgaga aagaaactgg tggacagcac cgacaaggcc gacctgcggc tgatctatct 840
ggccctggcc cacatgatca agttccgggg ccacttcctg atcgagggcg acctgaaccc 900
cgacaacagc gacgtggaca agctgttcat ccagctggtg cagacctaca accagctgtt 960
cgaggaaaac cccatcaacg ccagcggcgt ggacgccaag gccatcctgt ctgccagact 1020
gagcaagagc agacggctgg aaaatctgat cgcccagctg cccggcgaga agaagaatgg 1080
cctgttcggc aacctgattg ccctgagcct gggcctgacc cccaacttca agagcaactt 1140
cgacctggcc gaggatgcca aactgcagct gagcaaggac acctacgacg acgacctgga 1200
caacctgctg gcccagatcg gcgaccagta cgccgacctg tttctggccg ccaagaacct 1260
gtccgacgcc atcctgctga gcgacatcct gagagtgaac accgagatca ccaaggcccc 1320
cctgagcgcc tctatgatca agagatacga cgagcaccac caggacctga ccctgctgaa 1380
agctctcgtg cggcagcagc tgcctgagaa gtacaaagag attttcttcg accagagcaa 1440
gaacggctac gccggctaca ttgacggcgg agccagccag gaagagttct acaagttcat 1500
caagcccatc ctggaaaaga tggacggcac cgaggaactg ctcgtgaagc tgaacagaga 1560
ggacctgctg cggaagcagc ggaccttcga caacggcagc atcccccacc agatccacct 1620
gggagagctg cacgccattc tgcggcggca ggaagatttt tacccattcc tgaaggacaa 1680
ccgggaaaag atcgagaaga tcctgacctt ccgcatcccc tactacgtgg gccctctggc 1740
caggggaaac agcagattcg cctggatgac cagaaagagc gaggaaacca tcaccccctg 1800
gaacttcgag gaagtggtgg acaagggcgc ttccgcccag agcttcatcg agcggatgac 1860
caacttcgat aagaacctgc ccaacgagaa ggtgctgccc aagcacagcc tgctgtacga 1920
gtacttcacc gtgtataacg agctgaccaa agtgaaatac gtgaccgagg gaatgagaaa 1980
gcccgccttc ctgagcggcg agcagaaaaa ggccatcgtg gacctgctgt tcaagaccaa 2040
ccggaaagtg accgtgaagc agctgaaaga ggactacttc aagaaaatcg agtgcttcga 2100
ctccgtggaa atctccggcg tggaagatcg gttcaacgcc tccctgggca cataccacga 2160
tctgctgaaa attatcaagg acaaggactt cctggacaat gaggaaaacg aggacattct 2220
ggaagatatc gtgctgaccc tgacactgtt tgaggacaga gagatgatcg aggaacggct 2280
gaaaacctat gcccacctgt tcgacgacaa agtgatgaag cagctgaagc ggcggagata 2340
caccggctgg ggcaggctga gccggaagct gatcaacggc atccgggaca agcagtccgg 2400
caagacaatc ctggatttcc tgaagtccga cggcttcgcc aacagaaact tcatgcagct 2460
gatccacgac gacagcctga cctttaaaga ggacatccag aaagcccagg tgtccggcca 2520
gggcgatagc ctgcacgagc acattgccaa tctggccggc agccccgcca ttaagaaggg 2580
catcctgcag acagtgaagg tggtggacga gctcgtgaaa gtgatgggcc ggcacaagcc 2640
cgagaacatc gtgatcgaaa tggccagaga gaaccagacc acccagaagg gacagaagaa 2700
cagccgcgag agaatgaagc ggatcgaaga gggcatcaaa gagctgggca gccagatcct 2760
gaaagaacac cccgtggaaa acacccagct gcagaacgag aagctgtacc tgtactacct 2820
gcagaatggg cgggatatgt acgtggacca ggaactggac atcaaccggc tgtccgacta 2880
cgatgtggac catatcgtgc ctcagagctt tctgaaggac gactccatcg acaacaaggt 2940
gctgaccaga agcgacaaga accggggcaa gagcgacaac gtgccctccg aagaggtcgt 3000
gaagaagatg aagaactact ggcggcagct gctgaacgcc aagctgatta cccagagaaa 3060
gttcgacaat ctgaccaagg ccgagagagg cggcctgagc gaactggata aggccggctt 3120
catcaagaga cagctggtgg aaacccggca gatcacaaag cacgtggcac agatcctgga 3180
ctcccggatg aacactaagt acgacgagaa tgacaagctg atccgggaag tgaaagtgat 3240
caccctgaag tccaagctgg tgtccgattt ccggaaggat ttccagtttt acaaagtgcg 3300
cgagatcaac aactaccacc acgcccacga cgcctacctg aacgccgtcg tgggaaccgc 3360
cctgatcaaa aagtacccta agctggaaag cgagttcgtg tacggcgact acaaggtgta 3420
cgacgtgcgg aagatgatcg ccaagagcga gcaggaaatc ggcaaggcta ccgccaagta 3480
cttcttctac agcaacatca tgaacttttt caagaccgag attaccctgg ccaacggcga 3540
gatccggaag cggcctctga tcgagacaaa cggcgaaacc ggggagatcg tgtgggataa 3600
gggccgggat tttgccaccg tgcggaaagt gctgagcatg ccccaagtga atatcgtgaa 3660
aaagaccgag gtgcagacag gcggcttcag caaagagtct atcctgccca agaggaacag 3720
cgataagctg atcgccagaa agaaggactg ggaccctaag aagtacggcg gcttcgacag 3780
ccccaccgtg gcctattctg tgctggtggt ggccaaagtg gaaaagggca agtccaagaa 3840
actgaagagt gtgaaagagc tgctggggat caccatcatg gaaagaagca gcttcgagaa 3900
gaatcccatc gactttctgg aagccaaggg ctacaaagaa gtgaaaaagg acctgatcat 3960
caagctgcct aagtactccc tgttcgagct ggaaaacggc cggaagagaa tgctggcctc 4020
tgccggcgaa ctgcagaagg gaaacgaact ggccctgccc tccaaatatg tgaacttcct 4080
gtacctggcc agccactatg agaagctgaa gggctccccc gaggataatg agcagaaaca 4140
gctgtttgtg gaacagcaca agcactacct ggacgagatc atcgagcaga tcagcgagtt 4200
ctccaagaga gtgatcctgg ccgacgctaa tctggacaaa gtgctgtccg cctacaacaa 4260
gcaccgggat aagcccatca gagagcaggc cgagaatatc atccacctgt ttaccctgac 4320
caatctggga gcccctgccg ccttcaagta ctttgacacc accatcgacc ggaagaggta 4380
caccagcacc aaagaggtgc tggacgccac cctgatccac cagagcatca ccggcctgta 4440
cgagacacgg atcgacctgt ctcagctggg aggcgacagc cccaagaaga agagaaaggt 4500
ggaggccagc taaggatccg gcaagactgg ccccgcttgg caacgcaaca gtgagcccct 4560
ccctagtgtg tttggggatg tgactatgta ttcgtgtgtt ggccaacggg tcaacccgaa 4620
cagattgata cccgccttgg catttcctgt cagaatgtaa cgtcagttga tggtact 4677
<210> 20
<211> 3150
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 20
tctttcttgc gctatgacac ttccagcaaa aggtagggcg ggctgcgaga cggcttcccg 60
gcgctgcatg caacaccgat gatgcttcga ccccccgaag ctccttcggg gctgcatggg 120
cgctccgatg ccgctccagg gcgagcgctg tttaaatagc caggcccccg attgcaaaga 180
cattatagcg agctaccaaa gccatattca aacacctaga tcactaccac ttctacacag 240
gccactcgag cttgtgatcg cactccgcta agggggcgcc tcttcctctt cgtttcagtc 300
acaacccgca aacatgccta agaagaagag gaaggttaac acgattaaca tcgctaagaa 360
cgacttctct gacatcgaac tggctgctat cccgttcaac actctggctg accattacgg 420
tgagcgttta gctcgcgaac agttggccct tgagcatgag tcttacgaga tgggtgaagc 480
acgcttccgc aagatgtttg agcgtcaact taaagctggt gaggttgcgg ataacgctgc 540
cgccaagcct ctcatcacta ccctactccc taagatgatt gcacgcatca acgactggtt 600
tgaggaagtg aaagctaagc gcggcaagcg cccgacagcc ttccagttcc tgcaagaaat 660
caagccggaa gccgtagcgt acatcaccat taagaccact ctggcttgcc taaccagtgc 720
tgacaataca accgttcagg ctgtagcaag cgcaatcggt cgggccattg aggacgaggc 780
tcgcttcggt cgtatccgtg accttgaagc taagcacttc aagaaaaacg ttgaggaaca 840
actcaacaag cgcgtagggc acgtctacaa gaaagcattt atgcaagttg tcgaggctga 900
catgctctct aagggtctac tcggtggcga ggcgtggtct tcgtggcata aggaagactc 960
tattcatgta ggagtacgct gcatcgagat gctcattgag tcaaccggaa tggttagctt 1020
acaccgccaa aatgctggcg tagtaggtca agactctgag actatcgaac tcgcacctga 1080
atacgctgag gctatcgcaa cccgtgcagg tgcgctggct ggcatctctc cgatgttcca 1140
accttgcgta gttcctccta agccgtggac tggcattact ggtggtggct attgggctaa 1200
cggtcgtcgt cctctggcgc tggtgcgtac tcacagtaag aaagcactga tgcgctacga 1260
agacgtttac atgcctgagg tgtacaaagc gattaacatt gcgcaaaaca ccgcatggaa 1320
aatcaacaag aaagtcctag cggtcgccaa cgtaatcacc aagtggaagc attgtccggt 1380
cgaggacatc cctgcgattg agcgtgaaga actcccgatg aaaccggaag acatcgacat 1440
gaatcctgag gctctcaccg cgtggaaacg tgctgccgct gctgtgtacc gcaaggacaa 1500
ggctcgcaag tctcgccgta tcagccttga gttcatgctt gagcaagcca ataagtttgc 1560
taaccataag gccatctggt tcccttacaa catggactgg cgcggtcgtg tttacgctgt 1620
gtcaatgttc aacccgcaag gtaacgatat gaccaaagga ctgcttacgc tggcgaaagg 1680
taaaccaatc ggtaaggaag gttactactg gctgaaaatc cacggtgcaa actgtgcggg 1740
tgtcgacaag gttccgttcc ctgagcgcat caagttcatt gaggaaaacc acgagaacat 1800
catggcttgc gctaagtctc cactggagaa cacttggtgg gctgagcaag attctccgtt 1860
ctgcttcctt gcgttctgct ttgagtacgc tggggtacag caccacggcc tgagctataa 1920
ctgctccctt ccgctggcgt ttgacgggtc ttgctctggc atccagcact tctccgcgat 1980
gctccgagat gaggtaggtg gtcgcgcggt taacttgctt cctagtgaaa ccgttcagga 2040
catctacggg attgttgcta agaaagtcaa cgagattcta caagcagacg caatcaatgg 2100
gaccgataac gaagtagtta ccgtgaccga tgagaacact ggtgaaatct ctgagaaagt 2160
caagctgggc actaaggcac tggctggtca atggctggct tacggtgtta ctcgcagtgt 2220
gactaagcgt tcagtcatga cgctggctta cgggtccaaa gagttcggct tccgtcaaca 2280
agtgctggaa gataccattc agccagctat tgattccggc aagggtctga tgttcactca 2340
gccgaatcag gctgctggat acatggctaa gctgatttgg gaatctgtga gcgtgacggt 2400
ggtagctgcg gttgaagcaa tgaactggct taagtctgct gctaagctgc tggctgctga 2460
ggtcaaagat aagaagactg gagagattct tcgcaagcgt tgcgctgtgc attgggtaac 2520
tcctgatggt ttccctgtgt ggcaggaata caagaagcct attcagacgc gcttgaacct 2580
gatgttcctc ggtcagttcc gcttacagcc taccattaac accaacaaag atagcgagat 2640
tgatgcacac aaacaggagt ctggtatcgc tcctaacttt gtacacagcc aagacggtag 2700
ccaccttcgt aagactgtag tgtgggcaca cgagaagtac ggaatcgaat cttttgcact 2760
gattcacgac tccttcggta cgattccggc tgacgctgcg aacctgttca aagcagtgcg 2820
cgaaactatg gttgacacat atgagtcttg tgatgtactg gctgatttct acgaccagtt 2880
cgctgaccag ttgcacgagt ctcaattgga caaaatgcca gcacttccgg ctaaaggtaa 2940
cttgaacctc cgtgacatct tagagtcgga cttcgcgttc gcgtaaggat ccggcaagac 3000
tggccccgct tggcaacgca acagtgagcc cctccctagt gtgtttgggg atgtgactat 3060
gtattcgtgt gttggccaac gggtcaaccc gaacagattg atacccgcct tggcatttcc 3120
tgtcagaatg taacgtcagt tgatggtact 3150
<210> 21
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> modified_base
<222> (23)..(42)
<223> a, c, t, g, unknown or other
<400> 21
gaaattaata cgactcacta tannnnnnnn nnnnnnnnnn nngttttaga gctagaaata 60
gcaagttaaa ataaggctag tccgttatca acttgaaaaa gtggcaccga gtcggtgctt 120
ttttt 125
<210> 22
<211> 8452
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 22
tgcggtattt cacaccgcat caggtggcac ttttcgggga aatgtgcgcg gaacccctat 60
ttgtttattt ttctaaatac attcaaatat gtatccgctc atgagattat caaaaaggat 120
cttcacctag atccttttaa attaaaaatg aagttttaaa tcaatctaaa gtatatatga 180
gtaaacttgg tctgacagtt accaatgctt aatcagtgag gcacctatct cagcgatctg 240
tctatttcgt tcatccatag ttgcctgact ccccgtcgtg tagataacta cgatacggga 300
gggcttacca tctggcccca gtgctgcaat gataccgcga gacccacgct caccggctcc 360
agatttatca gcaataaacc agccagccgg aagggccgag cgcagaagtg gtcctgcaac 420
tttatccgcc tccatccagt ctattaattg ttgccgggaa gctagagtaa gtagttcgcc 480
agttaatagt ttgcgcaacg ttgttgccat tgctacaggc atcgtggtgt cacgctcgtc 540
gtttggtatg gcttcattca gctccggttc ccaacgatca aggcgagtta catgatcccc 600
catgttgtgc aaaaaagcgg ttagctcctt cggtcctccg atcgttgtca gaagtaagtt 660
ggccgcagtg ttatcactca tggttatggc agcactgcat aattctctta ctgtcatgcc 720
atccgtaaga tgcttttctg tgactggtga gtactcaacc aagtcattct gagaatagtg 780
tatgcggcga ccgagttgct cttgcccggc gtcaatacgg gataataccg cgccacatag 840
cagaacttta aaagtgctca tcattggaaa acgttcttcg gggcgaaaac tctcaaggat 900
cttaccgctg ttgagatcca gttcgatgta acccactcgt gcacccaact gatcttcagc 960
atcttttact ttcaccagcg tttctgggtg agcaaaaaca ggaaggcaaa atgccgcaaa 1020
aaagggaata agggcgacac ggaaatgttg aatactcata ctcttccttt ttcaatatta 1080
ttgaagcatt tatcagggtt attgtctcat gaccaaaatc ccttaacgtg agttttcgtt 1140
ccactgagcg tcagaccccg tagaaaagat caaaggatct tcttgagatc ctttttttct 1200
gcgcgtaatc tgctgcttgc aaacaaaaaa accaccgcta ccagcggtgg tttgtttgcc 1260
ggatcaagag ctaccaactc tttttccgaa ggtaactggc ttcagcagag cgcagatacc 1320
aaatactgtt cttctagtgt agccgtagtt aggccaccac ttcaagaact ctgtagcacc 1380
gcctacatac ctcgctctgc taatcctgtt accagtggct gttgccagtg gcgataagtc 1440
gtgtcttacc gggttggact caagacgata gttaccggat aaggcgcagc ggtcgggctg 1500
aacggggggt tcgtgcacac agcccagctt ggagcgaacg acctacaccg aactgagata 1560
cctacagcgt gagctatgag aaagcgccac gcttcccgaa gggagaaagg cggacaggta 1620
tccggtaagc ggcagggtcg gaacaggaga gcgcacgagg gagcttccag ggggaaacgc 1680
ctggtatctt tatagtcctg tcgggtttcg ccacctctga cttgagcgtc gatttttgtg 1740
atgctcgtca ggggggcgga gcctatggaa aaacgccagc aacgcggcct ttttacggtt 1800
cctggccttt tgctggcctt ttgctcacat gttctttcct gcgttatccc ctgattctgt 1860
ggataaccgt attaccgcct ttgagtgagc tgataccgct cgccgcagcc gaacgaccga 1920
gcgcagcgag tcagtgagcg aggaagcggt cgctgaggct tgacatgatt ggtgcgtatg 1980
tttgtatgaa gctacaggac tgatttggcg ggctatgagg gcgggggaag ctctggaagg 2040
gccgcgatgg ggcgcgcggc gtccagaagg cgccatacgg cccgctggcg gcacccatcc 2100
ggtataaaag cccgcgaccc cgaacggtga cctccacttt cagcgacaaa cgagcactta 2160
tacatacgcg actattctgc cgctatacat aaccactcag ctagcttaag atcccatcaa 2220
gcttgcatgc cgggcgcgcc agaaggagcg cagccaaacc aggatgatgt ttgatggggt 2280
atttgagcac ttgcaaccct tatccggaag ccccctggcc cacaaaggct aggcgccaat 2340
gcaagcagtt cgcatgcagc ccctggagcg gtgccctcct gataaaccgg ccagggggcc 2400
tatgttcttt acttttttac aagagaagtc actcaacatc ttaaaatggc caggtgagtc 2460
gacgagcaag cccggcggat caggcagcgt gcttgcagat ttgacttgca acgcccgcat 2520
tgtgtcgacg aaggcttttg gctcctctgt cgctgtctca agcagcatct aaccctgcgt 2580
cgccgtttcc atttgcagga gattcgaggt accatgtacc catacgatgt tccagattac 2640
gcttcgccga agaaaaagcg caaggtcgaa gcgtccgaca agaagtacag catcggcctg 2700
gacatcggca ccaactctgt gggctgggcc gtgatcaccg acgagtacaa ggtgcccagc 2760
aagaaattca aggtgctggg caacaccgac cggcacagca tcaagaagaa cctgatcgga 2820
gccctgctgt tcgacagcgg cgaaacagcc gaggccaccc ggctgaagag aaccgccaga 2880
agaagataca ccagacggaa gaaccggatc tgctatctgc aagagatctt cagcaacgag 2940
atggccaagg tggacgacag cttcttccac agactggaag agtccttcct ggtggaagag 3000
gataagaagc acgagcggca ccccatcttc ggcaacatcg tggacgaggt ggcctaccac 3060
gagaagtacc ccaccatcta ccacctgaga aagaaactgg tggacagcac cgacaaggcc 3120
gacctgcggc tgatctatct ggccctggcc cacatgatca agttccgggg ccacttcctg 3180
atcgagggcg acctgaaccc cgacaacagc gacgtggaca agctgttcat ccagctggtg 3240
cagacctaca accagctgtt cgaggaaaac cccatcaacg ccagcggcgt ggacgccaag 3300
gccatcctgt ctgccagact gagcaagagc agacggctgg aaaatctgat cgcccagctg 3360
cccggcgaga agaagaatgg cctgttcggc aacctgattg ccctgagcct gggcctgacc 3420
cccaacttca agagcaactt cgacctggcc gaggatgcca aactgcagct gagcaaggac 3480
acctacgacg acgacctgga caacctgctg gcccagatcg gcgaccagta cgccgacctg 3540
tttctggccg ccaagaacct gtccgacgcc atcctgctga gcgacatcct gagagtgaac 3600
accgagatca ccaaggcccc cctgagcgcc tctatgatca agagatacga cgagcaccac 3660
caggacctga ccctgctgaa agctctcgtg cggcagcagc tgcctgagaa gtacaaagag 3720
attttcttcg accagagcaa gaacggctac gccggctaca ttgacggcgg agccagccag 3780
gaagagttct acaagttcat caagcccatc ctggaaaaga tggacggcac cgaggaactg 3840
ctcgtgaagc tgaacagaga ggacctgctg cggaagcagc ggaccttcga caacggcagc 3900
atcccccacc agatccacct gggagagctg cacgccattc tgcggcggca ggaagatttt 3960
tacccattcc tgaaggacaa ccgggaaaag atcgagaaga tcctgacctt ccgcatcccc 4020
tactacgtgg gccctctggc caggggaaac agcagattcg cctggatgac cagaaagagc 4080
gaggaaacca tcaccccctg gaacttcgag gaagtggtgg acaagggcgc ttccgcccag 4140
agcttcatcg agcggatgac caacttcgat aagaacctgc ccaacgagaa ggtgctgccc 4200
aagcacagcc tgctgtacga gtacttcacc gtgtataacg agctgaccaa agtgaaatac 4260
gtgaccgagg gaatgagaaa gcccgccttc ctgagcggcg agcagaaaaa ggccatcgtg 4320
gacctgctgt tcaagaccaa ccggaaagtg accgtgaagc agctgaaaga ggactacttc 4380
aagaaaatcg agtgcttcga ctccgtggaa atctccggcg tggaagatcg gttcaacgcc 4440
tccctgggca cataccacga tctgctgaaa attatcaagg acaaggactt cctggacaat 4500
gaggaaaacg aggacattct ggaagatatc gtgctgaccc tgacactgtt tgaggacaga 4560
gagatgatcg aggaacggct gaaaacctat gcccacctgt tcgacgacaa agtgatgaag 4620
cagctgaagc ggcggagata caccggctgg ggcaggctga gccggaagct gatcaacggc 4680
atccgggaca agcagtccgg caagacaatc ctggatttcc tgaagtccga cggcttcgcc 4740
aacagaaact tcatgcagct gatccacgac gacagcctga cctttaaaga ggacatccag 4800
aaagcccagg tgtccggcca gggcgatagc ctgcacgagc acattgccaa tctggccggc 4860
agccccgcca ttaagaaggg catcctgcag acagtgaagg tggtggacga gctcgtgaaa 4920
gtgatgggcc ggcacaagcc cgagaacatc gtgatcgaaa tggccagaga gaaccagacc 4980
acccagaagg gacagaagaa cagccgcgag agaatgaagc ggatcgaaga gggcatcaaa 5040
gagctgggca gccagatcct gaaagaacac cccgtggaaa acacccagct gcagaacgag 5100
aagctgtacc tgtactacct gcagaatggg cgggatatgt acgtggacca ggaactggac 5160
atcaaccggc tgtccgacta cgatgtggac catatcgtgc ctcagagctt tctgaaggac 5220
gactccatcg acaacaaggt gctgaccaga agcgacaaga accggggcaa gagcgacaac 5280
gtgccctccg aagaggtcgt gaagaagatg aagaactact ggcggcagct gctgaacgcc 5340
aagctgatta cccagagaaa gttcgacaat ctgaccaagg ccgagagagg cggcctgagc 5400
gaactggata aggccggctt catcaagaga cagctggtgg aaacccggca gatcacaaag 5460
cacgtggcac agatcctgga ctcccggatg aacactaagt acgacgagaa tgacaagctg 5520
atccgggaag tgaaagtgat caccctgaag tccaagctgg tgtccgattt ccggaaggat 5580
ttccagtttt acaaagtgcg cgagatcaac aactaccacc acgcccacga cgcctacctg 5640
aacgccgtcg tgggaaccgc cctgatcaaa aagtacccta agctggaaag cgagttcgtg 5700
tacggcgact acaaggtgta cgacgtgcgg aagatgatcg ccaagagcga gcaggaaatc 5760
ggcaaggcta ccgccaagta cttcttctac agcaacatca tgaacttttt caagaccgag 5820
attaccctgg ccaacggcga gatccggaag cggcctctga tcgagacaaa cggcgaaacc 5880
ggggagatcg tgtgggataa gggccgggat tttgccaccg tgcggaaagt gctgagcatg 5940
ccccaagtga atatcgtgaa aaagaccgag gtgcagacag gcggcttcag caaagagtct 6000
atcctgccca agaggaacag cgataagctg atcgccagaa agaaggactg ggaccctaag 6060
aagtacggcg gcttcgacag ccccaccgtg gcctattctg tgctggtggt ggccaaagtg 6120
gaaaagggca agtccaagaa actgaagagt gtgaaagagc tgctggggat caccatcatg 6180
gaaagaagca gcttcgagaa gaatcccatc gactttctgg aagccaaggg ctacaaagaa 6240
gtgaaaaagg acctgatcat caagctgcct aagtactccc tgttcgagct ggaaaacggc 6300
cggaagagaa tgctggcctc tgccggcgaa ctgcagaagg gaaacgaact ggccctgccc 6360
tccaaatatg tgaacttcct gtacctggcc agccactatg agaagctgaa gggctccccc 6420
gaggataatg agcagaaaca gctgtttgtg gaacagcaca agcactacct ggacgagatc 6480
atcgagcaga tcagcgagtt ctccaagaga gtgatcctgg ccgacgctaa tctggacaaa 6540
gtgctgtccg cctacaacaa gcaccgggat aagcccatca gagagcaggc cgagaatatc 6600
atccacctgt ttaccctgac caatctggga gcccctgccg ccttcaagta ctttgacacc 6660
accatcgacc ggaagaggta caccagcacc aaagaggtgc tggacgccac cctgatccac 6720
cagagcatca ccggcctgta cgagacacgg atcgacctgt ctcagctggg aggcgacagc 6780
cccaagaaga agagaaaggt ggaggccagc taacatatga ttcgaatgtc tttcttgcgc 6840
tatgacactt ccagcaaaag gtagggcggg ctgcgagacg gcttcccggc gctgcatgca 6900
acaccgatga tgcttcgacc ccccgaagct ccttcggggc tgcatgggcg ctccgatgcc 6960
gctccagggc gagcgctgtt taaatagcca ggcccccgat tgcaaagaca ttatagcgag 7020
ctaccaaagc catattcaaa cacctagatc actaccactt ctacacaggc cactcgagct 7080
tgtgatcgca ctccgctaag ggggcgcctc ttcctcttcg tttcagtcac aacccgcaaa 7140
catgacacaa gaatccctgt tacttctcga ccgtattgat tcggatgatt cctacgcgag 7200
cctgcggaac gaccaggaat tctgggaggt gagtcgacga gcaagcccgg cggatcaggc 7260
agcgtgcttg cagatttgac ttgcaacgcc cgcattgtgt cgacgaaggc ttttggctcc 7320
tctgtcgctg tctcaagcag catctaaccc tgcgtcgccg tttccatttg cagccgctgg 7380
cccgccgagc cctggaggag ctcgggctgc cggtgccgcc ggtgctgcgg gtgcccggcg 7440
agagcaccaa ccccgtactg gtcggcgagc ccggcccggt gatcaagctg ttcggcgagc 7500
actggtgcgg tccggagagc ctcgcgtcgg agtcggaggc gtacgcggtc ctggcggacg 7560
ccccggtgcc ggtgccccgc ctcctcggcc gcggcgagct gcggcccggc accggagcct 7620
ggccgtggcc ctacctggtg atgagccgga tgaccggcac cacctggcgg tccgcgatgg 7680
acggcacgac cgaccggaac gcgctgctcg ccctggcccg cgaactcggc cgggtgctcg 7740
gccggctgca cagggtgccg ctgaccggga acaccgtgct caccccccat tccgaggtct 7800
tcccggaact gctgcgggaa cgccgcgcgg cgaccgtcga ggaccaccgc gggtggggct 7860
acctctcgcc ccggctgctg gaccgcctgg aggactggct gccggacgtg gacacgctgc 7920
tggccggccg cgaaccccgg ttcgtccacg gcgacctgca cgggaccaac atcttcgtgg 7980
acctggccgc gaccgaggtc accgggatcg tcgacttcac cgacgtctat gcgggagact 8040
cccgctacag cctggtgcaa ctgcatctca acgccttccg gggcgaccgc gagatcctgg 8100
ccgcgctgct cgacggggcg cagtggaagc ggaccgagga cttcgcccgc gaactgctcg 8160
ccttcacctt cctgcacgac ttcgaggtgt tcgaggagac cccgctggat ctctccggct 8220
tcaccgatcc ggaggaactg gcgcagttcc tctgggggcc gccggacacc gcccccggcg 8280
cctgataagg atccggcaag actggccccg cttggcaacg caacagtgag cccctcccta 8340
gtgtgtttgg ggatgtgact atgtattcgt gtgttggcca acgggtcaac ccgaacagat 8400
tgatacccgc cttggcattt cctgtcagaa tgtaacgtca gttgatggta ct 8452
<210> 23
<211> 102
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 23
gttttagagc tatgctgttt tgaatggtcc caaaacggaa gggcctgagt ccgagcagaa 60
gaagaagttt tagagctatg ctgttttgaa tggtcccaaa ac 102
<210> 24
<211> 100
<212> DNA
<213> Homo sapiens
<400> 24
cggaggacaa agtacaaacg gcagaagctg gaggaggaag ggcctgagtc cgagcagaag 60
aagaagggct cccatcacat caaccggtgg cgcattgcca 100
<210> 25
<211> 50
<212> DNA
<213> Homo sapiens
<400> 25
agctggagga ggaagggcct gagtccgagc agaagaagaa gggctcccac 50
<210> 26
<211> 30
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 26
gaguccgagc agaagaagaa guuuuagagc 30
<210> 27
<211> 49
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 27
agctggagga ggaagggcct gagtccgagc agaagagaag ggctcccat 49
<210> 28
<211> 53
<212> DNA
<213> Homo sapiens
<400> 28
ctggaggagg aagggcctga gtccgagcag aagaagaagg gctcccatca cat 53
<210> 29
<211> 52
<212> DNA
<213> Homo sapiens
<400> 29
ctggaggagg aagggcctga gtccgagcag aagagaaggg ctcccatcac at 52
<210> 30
<211> 54
<212> DNA
<213> Homo sapiens
<400> 30
ctggaggagg aagggcctga gtccgagcag aagaaagaag ggctcccatc acat 54
<210> 31
<211> 50
<212> DNA
<213> Homo sapiens
<400> 31
ctggaggagg aagggcctga gtccgagcag aagaagggct cccatcacat 50
<210> 32
<211> 47
<212> DNA
<213> Homo sapiens
<400> 32
ctggaggagg aagggcctga gcccgagcag aagggctccc atcacat 47
<210> 33
<211> 48
<212> DNA
<213> Homo sapiens
<400> 33
ctggaggagg aagggcctga gtccgagcag aagaagaagg gctcccat 48
<210> 34
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 34
gaguccgagc agaagaagau 20
<210> 35
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 35
gaguccgagc agaagaagua 20
<210> 36
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 36
gaguccgagc agaagaacaa 20
<210> 37
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 37
gaguccgagc agaagaugaa 20
<210> 38
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 38
gaguccgagc agaaguagaa 20
<210> 39
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 39
gaguccgagc agaugaagaa 20
<210> 40
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 40
gaguccgagc acaagaagaa 20
<210> 41
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 41
gaguccgagg agaagaagaa 20
<210> 42
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 42
gaguccgugc agaagaagaa 20
<210> 43
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 43
gagucggagc agaagaagaa 20
<210> 44
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 44
gagaccgagc agaagaagaa 20
<210> 45
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 45
aatgacaagc ttgctagcgg tggg 24
<210> 46
<211> 39
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 46
aaaacggaag ggcctgagtc cgagcagaag aagaagttt 39
<210> 47
<211> 39
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 47
aaacaggggc cgagattggg tgttcagggc agaggtttt 39
<210> 48
<211> 38
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 48
aaaacggaag ggcctgagtc cgagcagaag aagaagtt 38
<210> 49
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 49
aacggaggga ggggcacaga tgagaaactc agggttttag 40
<210> 50
<211> 38
<212> DNA
<213> Homo sapiens
<400> 50
agcccttctt cttctgctcg gactcaggcc cttcctcc 38
<210> 51
<211> 40
<212> DNA
<213> Homo sapiens
<400> 51
cagggaggga ggggcacaga tgagaaactc aggaggcccc 40
<210> 52
<211> 80
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 52
ggcaatgcgc caccggttga tgtgatggga gcccttctag gaggccccca gagcagccac 60
tggggcctca acactcaggc 80
<210> 53
<211> 33
<212> DNA
<213> Homo sapiens
<400> 53
catcgatgtc ctccccattg gcctgcttcg tgg 33
<210> 54
<211> 33
<212> DNA
<213> Homo sapiens
<400> 54
ttcgtggcaa tgcgccaccg gttgatgtga tgg 33
<210> 55
<211> 33
<212> DNA
<213> Homo sapiens
<400> 55
tcgtggcaat gcgccaccgg ttgatgtgat ggg 33
<210> 56
<211> 33
<212> DNA
<213> Homo sapiens
<400> 56
tccagcttct gccgtttgta ctttgtcctc cgg 33
<210> 57
<211> 33
<212> DNA
<213> Homo sapiens
<400> 57
ggagggaggg gcacagatga gaaactcagg agg 33
<210> 58
<211> 33
<212> DNA
<213> Homo sapiens
<400> 58
aggggccgag attgggtgtt cagggcagag agg 33
<210> 59
<211> 33
<212> DNA
<213> Mus musculus
<400> 59
caagcactga gtgccattag ctaaatgcat agg 33
<210> 60
<211> 33
<212> DNA
<213> Mus musculus
<400> 60
aatgcatagg gtaccaccca caggtgccag ggg 33
<210> 61
<211> 33
<212> DNA
<213> Mus musculus
<400> 61
acacacatgg gaaagcctct gggccaggaa agg 33
<210> 62
<211> 37
<212> DNA
<213> Homo sapiens
<400> 62
ggaggaggta gtatacagaa acacagagaa gtagaat 37
<210> 63
<211> 37
<212> DNA
<213> Homo sapiens
<400> 63
agaatgtaga ggagtcacag aaactcagca ctagaaa 37
<210> 64
<211> 98
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 64
ggacgaaaca ccggaaccat tcaaaacagc atagcaagtt aaaataaggc tagtccgtta 60
tcaacttgaa aaagtggcac cgagtcggtg cttttttt 98
<210> 65
<211> 186
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 65
ggacgaaaca ccggtagtat taagtattgt tttatggctg ataaatttct ttgaatttct 60
ccttgattat ttgttataaa agttataaaa taatcttgtt ggaaccattc aaaacagcat 120
agcaagttaa aataaggcta gtccgttatc aacttgaaaa agtggcaccg agtcggtgct 180
tttttt 186
<210> 66
<211> 95
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 66
gggttttaga gctatgctgt tttgaatggt cccaaaacgg gtcttcgaga agacgtttta 60
gagctatgct gttttgaatg gtcccaaaac ttttt 95
<210> 67
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<220>
<221> modified_base
<222> (5)..(34)
<223> a, c, t, g, unknown or other
<400> 67
aaacnnnnnn nnnnnnnnnn nnnnnnnnnn nnnngt 36
<210> 68
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<220>
<221> modified_base
<222> (7)..(36)
<223> a, c, t, g, unknown or other
<400> 68
taaaacnnnn nnnnnnnnnn nnnnnnnnnn nnnnnn 36
<210> 69
<211> 84
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 69
gtggaaagga cgaaacaccg ggtcttcgag aagacctgtt ttagagctag aaatagcaag 60
ttaaaataag gctagtccgt tttt 84
<210> 70
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<220>
<221> modified_base
<222> (6)..(24)
<223> a, c, t, g, unknown or other
<400> 70
caccgnnnnn nnnnnnnnnn nnnn 24
<210> 71
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<220>
<221> modified_base
<222> (5)..(23)
<223> a, c, t, g, unknown or other
<400> 71
aaacnnnnnn nnnnnnnnnn nnnc 24
<210> 72
<211> 46
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<220>
<221> modified_base
<222> (1)..(19)
<223> a, c, u, g, unknown or other
<400> 72
nnnnnnnnnn nnnnnnnnng uuauuguacu cucaagauuu auuuuu 46
<210> 73
<211> 91
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 73
guuacuuaaa ucuugcagaa gcuacaaaga uaaggcuuca ugccgaaauc aacacccugu 60
cauuuuaugg caggguguuu ucguuauuua a 91
<210> 74
<211> 70
<212> DNA
<213> Homo sapiens
<400> 74
ttttctagtg ctgagtttct gtgactcctc tacattctac ttctctgtgt ttctgtatac 60
tacctcctcc 70
<210> 75
<211> 122
<212> DNA
<213> Homo sapiens
<400> 75
ggaggaaggg cctgagtccg agcagaagaa gaagggctcc catcacatca accggtggcg 60
cattgccacg aagcaggcca atggggagga catcgatgtc acctccaatg actagggtgg 120
gc 122
<210> 76
<211> 48
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<220>
<221> modified_base
<222> (3)..(32)
<223> a, c, u, g, unknown or other
<400> 76
acnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnguuuuaga gcuaugcu 48
<210> 77
<211> 67
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<220>
<221> source
<223> /note="Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide"
<400> 77
agcauagcaa guuaaaauaa ggctaguccg uuaucaacuu gaaaaagugg caccgagucg 60
gugcuuu 67
<210> 78
<211> 62
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, u, g, unknown or other
<400> 78
nnnnnnnnnn nnnnnnnnnn guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cg 62
<210> 79
<211> 73
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 79
tgaatggtcc caaaacggaa gggcctgagt ccgagcagaa gaagaagttt tagagctatg 60
ctgttttgaa tgg 73
<210> 80
<211> 69
<212> DNA
<213> Homo sapiens
<400> 80
ctggtcttcc acctctctgc cctgaacacc caatctcggc ccctctcgcc accctcctgc 60
atttctgtt 69
<210> 81
<211> 138
<212> DNA
<213> Mus musculus
<400> 81
acccaagcac tgagtgccat tagctaaatg catagggtac cacccacagg tgccaggggc 60
ctttcccaaa gttcccagcc ccttctccaa cctttcctgg cccagaggct ttcccatgtg 120
tgtggctgga ccctttga 138
<210> 82
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
primer"
<400> 82
aaaaccaccc ttctctctgg c 21
<210> 83
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
primer"
<400> 83
ggagattgga gacacggaga g 21
<210> 84
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
primer"
<400> 84
ctggaaagcc aatgcctgac 20
<210> 85
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
primer"
<400> 85
ggcagcaaac tccttgtcct 20
<210> 86
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
primer"
<400> 86
gtgctttgca gaggcctacc 20
<210> 87
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
primer"
<400> 87
cctggagcgc atgcagtagt 20
<210> 88
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
primer"
<400> 88
accttctgtg tttccaccat tc 22
<210> 89
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
primer"
<400> 89
ttggggagtg cacagacttc 20
<210> 90
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
primer"
<400> 90
ggctccctgg gttcaaagta 20
<210> 91
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
primer"
<400> 91
agaggggtct ggatgtcgta a 21
<210> 92
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
probe"
<400> 92
tagctctaaa acttcttctt ctgctcggac 30
<210> 93
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
probe"
<400> 93
ctagccttat tttaacttgc tatgctgttt 30
<210> 94
<211> 99
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, u, g, unknown or other
<400> 94
nnnnnnnnnn nnnnnnnnnn guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcuuu 99
<210> 95
<211> 12
<212> DNA
<213> Homo sapiens
<400> 95
tagcgggtaa gc 12
<210> 96
<211> 12
<212> DNA
<213> Homo sapiens
<400> 96
tcggtgacat gt 12
<210> 97
<211> 12
<212> DNA
<213> Homo sapiens
<400> 97
actccccgta gg 12
<210> 98
<211> 12
<212> DNA
<213> Homo sapiens
<400> 98
actgcgtgtt aa 12
<210> 99
<211> 12
<212> DNA
<213> Homo sapiens
<400> 99
acgtcgcctg at 12
<210> 100
<211> 12
<212> DNA
<213> Homo sapiens
<400> 100
taggtcgacc ag 12
<210> 101
<211> 12
<212> DNA
<213> Homo sapiens
<400> 101
ggcgttaatg at 12
<210> 102
<211> 12
<212> DNA
<213> Homo sapiens
<400> 102
tgtcgcatgt ta 12
<210> 103
<211> 12
<212> DNA
<213> Homo sapiens
<400> 103
atggaaacgc at 12
<210> 104
<211> 12
<212> DNA
<213> Homo sapiens
<400> 104
gccgaattcc tc 12
<210> 105
<211> 12
<212> DNA
<213> Homo sapiens
<400> 105
gcatggtacg ga 12
<210> 106
<211> 12
<212> DNA
<213> Homo sapiens
<400> 106
cggtactctt ac 12
<210> 107
<211> 12
<212> DNA
<213> Homo sapiens
<400> 107
gcctgtgccg ta 12
<210> 108
<211> 12
<212> DNA
<213> Homo sapiens
<400> 108
tacggtaagt cg 12
<210> 109
<211> 12
<212> DNA
<213> Homo sapiens
<400> 109
cacgaaatta cc 12
<210> 110
<211> 12
<212> DNA
<213> Homo sapiens
<400> 110
aaccaagata cg 12
<210> 111
<211> 12
<212> DNA
<213> Homo sapiens
<400> 111
gagtcgatac gc 12
<210> 112
<211> 12
<212> DNA
<213> Homo sapiens
<400> 112
gtctcacgat cg 12
<210> 113
<211> 12
<212> DNA
<213> Homo sapiens
<400> 113
tcgtcgggtg ca 12
<210> 114
<211> 12
<212> DNA
<213> Homo sapiens
<400> 114
actccgtagt ga 12
<210> 115
<211> 12
<212> DNA
<213> Homo sapiens
<400> 115
caggacgtcc gt 12
<210> 116
<211> 12
<212> DNA
<213> Homo sapiens
<400> 116
tcgtatccct ac 12
<210> 117
<211> 12
<212> DNA
<213> Homo sapiens
<400> 117
tttcaaggcc gg 12
<210> 118
<211> 12
<212> DNA
<213> Homo sapiens
<400> 118
cgccggtgga at 12
<210> 119
<211> 12
<212> DNA
<213> Homo sapiens
<400> 119
gaacccgtcc ta 12
<210> 120
<211> 12
<212> DNA
<213> Homo sapiens
<400> 120
gattcatcag cg 12
<210> 121
<211> 12
<212> DNA
<213> Homo sapiens
<400> 121
acaccggtct tc 12
<210> 122
<211> 12
<212> DNA
<213> Homo sapiens
<400> 122
atcgtgccct aa 12
<210> 123
<211> 12
<212> DNA
<213> Homo sapiens
<400> 123
gcgtcaatgt tc 12
<210> 124
<211> 12
<212> DNA
<213> Homo sapiens
<400> 124
ctccgtatct cg 12
<210> 125
<211> 12
<212> DNA
<213> Homo sapiens
<400> 125
ccgattcctt cg 12
<210> 126
<211> 12
<212> DNA
<213> Homo sapiens
<400> 126
tgcgcctcca gt 12
<210> 127
<211> 12
<212> DNA
<213> Homo sapiens
<400> 127
taacgtcgga gc 12
<210> 128
<211> 12
<212> DNA
<213> Homo sapiens
<400> 128
aaggtcgccc at 12
<210> 129
<211> 12
<212> DNA
<213> Homo sapiens
<400> 129
gtcggggact at 12
<210> 130
<211> 12
<212> DNA
<213> Homo sapiens
<400> 130
ttcgagcgat tt 12
<210> 131
<211> 12
<212> DNA
<213> Homo sapiens
<400> 131
tgagtcgtcg ag 12
<210> 132
<211> 12
<212> DNA
<213> Homo sapiens
<400> 132
tttacgcaga gg 12
<210> 133
<211> 12
<212> DNA
<213> Homo sapiens
<400> 133
aggaagtatc gc 12
<210> 134
<211> 12
<212> DNA
<213> Homo sapiens
<400> 134
actcgatacc at 12
<210> 135
<211> 12
<212> DNA
<213> Homo sapiens
<400> 135
cgctacatag ca 12
<210> 136
<211> 12
<212> DNA
<213> Homo sapiens
<400> 136
ttcataaccg gc 12
<210> 137
<211> 12
<212> DNA
<213> Homo sapiens
<400> 137
ccaaacggtt aa 12
<210> 138
<211> 12
<212> DNA
<213> Homo sapiens
<400> 138
cgattccttc gt 12
<210> 139
<211> 12
<212> DNA
<213> Homo sapiens
<400> 139
cgtcatgaat aa 12
<210> 140
<211> 12
<212> DNA
<213> Homo sapiens
<400> 140
agtggcgatg ac 12
<210> 141
<211> 12
<212> DNA
<213> Homo sapiens
<400> 141
cccctacggc ac 12
<210> 142
<211> 12
<212> DNA
<213> Homo sapiens
<400> 142
gccaacccgc ac 12
<210> 143
<211> 12
<212> DNA
<213> Homo sapiens
<400> 143
tgggacaccg gt 12
<210> 144
<211> 12
<212> DNA
<213> Homo sapiens
<400> 144
ttgactgcgg cg 12
<210> 145
<211> 12
<212> DNA
<213> Homo sapiens
<400> 145
actatgcgta gg 12
<210> 146
<211> 12
<212> DNA
<213> Homo sapiens
<400> 146
tcacccaaag cg 12
<210> 147
<211> 12
<212> DNA
<213> Homo sapiens
<400> 147
gcaggacgtc cg 12
<210> 148
<211> 12
<212> DNA
<213> Homo sapiens
<400> 148
acaccgaaaa cg 12
<210> 149
<211> 12
<212> DNA
<213> Homo sapiens
<400> 149
cggtgtattg ag 12
<210> 150
<211> 12
<212> DNA
<213> Homo sapiens
<400> 150
cacgaggtat gc 12
<210> 151
<211> 12
<212> DNA
<213> Homo sapiens
<400> 151
taaagcgacc cg 12
<210> 152
<211> 12
<212> DNA
<213> Homo sapiens
<400> 152
cttagtcggc ca 12
<210> 153
<211> 12
<212> DNA
<213> Homo sapiens
<400> 153
cgaaaacgtg gc 12
<210> 154
<211> 12
<212> DNA
<213> Homo sapiens
<400> 154
cgtgccctga ac 12
<210> 155
<211> 12
<212> DNA
<213> Homo sapiens
<400> 155
tttaccatcg aa 12
<210> 156
<211> 12
<212> DNA
<213> Homo sapiens
<400> 156
cgtagccatg tt 12
<210> 157
<211> 12
<212> DNA
<213> Homo sapiens
<400> 157
cccaaacggt ta 12
<210> 158
<211> 12
<212> DNA
<213> Homo sapiens
<400> 158
gcgttatcag aa 12
<210> 159
<211> 12
<212> DNA
<213> Homo sapiens
<400> 159
tcgatggtaa ac 12
<210> 160
<211> 12
<212> DNA
<213> Homo sapiens
<400> 160
cgactttttg ca 12
<210> 161
<211> 12
<212> DNA
<213> Homo sapiens
<400> 161
tcgacgactc ac 12
<210> 162
<211> 12
<212> DNA
<213> Homo sapiens
<400> 162
acgcgtcaga ta 12
<210> 163
<211> 12
<212> DNA
<213> Homo sapiens
<400> 163
cgtacggcac ag 12
<210> 164
<211> 12
<212> DNA
<213> Homo sapiens
<400> 164
ctatgccgtg ca 12
<210> 165
<211> 12
<212> DNA
<213> Homo sapiens
<400> 165
cgcgtcagat at 12
<210> 166
<211> 12
<212> DNA
<213> Homo sapiens
<400> 166
aagatcggta gc 12
<210> 167
<211> 12
<212> DNA
<213> Homo sapiens
<400> 167
cttcgcaagg ag 12
<210> 168
<211> 12
<212> DNA
<213> Homo sapiens
<400> 168
gtcgtggact ac 12
<210> 169
<211> 12
<212> DNA
<213> Homo sapiens
<400> 169
ggtcgtcatc aa 12
<210> 170
<211> 12
<212> DNA
<213> Homo sapiens
<400> 170
gttaacagcg tg 12
<210> 171
<211> 12
<212> DNA
<213> Homo sapiens
<400> 171
tagctaaccg tt 12
<210> 172
<211> 12
<212> DNA
<213> Homo sapiens
<400> 172
agtaaaggcg ct 12
<210> 173
<211> 12
<212> DNA
<213> Homo sapiens
<400> 173
ggtaatttcg tg 12
<210> 174
<211> 15
<212> DNA
<213> Homo sapiens
<400> 174
cagaagaaga agggc 15
<210> 175
<211> 51
<212> DNA
<213> Homo sapiens
<400> 175
ccaatgggga ggacatcgat gtcacctcca atgactaggg tggtgggcaa c 51
<210> 176
<211> 15
<212> DNA
<213> Homo sapiens
<400> 176
ctctggccac tccct 15
<210> 177
<211> 52
<212> DNA
<213> Homo sapiens
<400> 177
acatcgatgt cacctccaat gacaagcttg ctagcggtgg gcaaccacaa ac 52
<210> 178
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<220>
<221> modified_base
<222> (6)..(25)
<223> a, c, t, g, unknown or other
<400> 178
caccgnnnnn nnnnnnnnnn nnnnn 25
<210> 179
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<220>
<221> modified_base
<222> (5)..(24)
<223> a, c, t, g, unknown or other
<400> 179
aaacnnnnnn nnnnnnnnnn nnnnc 25
<210> 180
<211> 54
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 180
aacaccgggt cttcgagaag acctgtttta gagctagaaa tagcaagtta aaat 54
<210> 181
<211> 54
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 181
caaaacgggt cttcgagaag acgttttaga gctatgctgt tttgaatggt ccca 54
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| КОНСТРУИРОВАНИЕ СИСТЕМ, СПОСОБЫ И ОПТИМИЗИРОВАННЫЕ НАПРАВЛЯЮЩИЕ КОМПОЗИЦИИ ДЛЯ МАНИПУЛЯЦИИ С ПОСЛЕДОВАТЕЛЬНОСТЯМИ | 2013 |
|
RU2796017C2 |
| КОМПОНЕНТЫ СИСТЕМЫ CRISPR-CAS, СПОСОБЫ И КОМПОЗИЦИИ ДЛЯ МАНИПУЛЯЦИИ С ПОСЛЕДОВАТЕЛЬНОСТЯМИ | 2013 |
|
RU2796549C2 |
| МУТАЦИИ ФЕРМЕНТА CRISPR, УМЕНЬШАЮЩИЕ НЕЦЕЛЕВЫЕ ЭФФЕКТЫ | 2016 |
|
RU2752834C2 |
| ДОСТАВКА, КОНСТРУИРОВАНИЕ И ОПТИМИЗАЦИЯ СИСТЕМ, СПОСОБОВ И КОМПОЗИЦИЙ ДЛЯ МАНИПУЛЯЦИИ С ПОСЛЕДОВАТЕЛЬНОСТЯМИ И ПРИМЕНЕНИЯ В ТЕРАПИИ | 2013 |
|
RU2721275C2 |
| НОВЫЕ ФЕРМЕНТЫ И СИСТЕМЫ CRISPR | 2016 |
|
RU2777988C2 |
| ДНК-СОДЕРЖАЩИЕ ПОЛИНУКЛЕОТИДЫ И РУКОВОДЯЩИЕ ПОСЛЕДОВАТЕЛЬНОСТИ ДЛЯ СИСТЕМ CRISPR ТИПА V И СПОСОБЫ ИХ ПОЛУЧЕНИЯ И ПРИМЕНЕНИЯ | 2021 |
|
RU2832314C1 |
| НОВЫЕ ФЕРМЕНТЫ CRISPR И СИСТЕМЫ | 2016 |
|
RU2771826C2 |
| ДОСТАВКА, ПРИМЕНЕНИЕ И ПРИМЕНЕНИЯ В ТЕРАПИИ СИСТЕМ CRISPR-CAS И КОМПОЗИЦИЙ ДЛЯ ЦЕЛЕНАПРАВЛЕННОГО ВОЗДЕЙСТВИЯ НА НАРУШЕНИЯ И ЗАБОЛЕВАНИЯ С ИСПОЛЬЗОВАНИЕМ ВИРУСНЫХ КОМПОНЕНТОВ | 2014 |
|
RU2716421C2 |
| ДОСТАВКА, КОНСТРУИРОВАНИЕ И ОПТИМИЗАЦИЯ СИСТЕМ, СПОСОБЫ И КОМПОЗИЦИИ ДЛЯ ЦЕЛЕНАПРАВЛЕННОГО ВОЗДЕЙСТВИЯ И МОДЕЛИРОВАНИЯ ЗАБОЛЕВАНИЙ И НАРУШЕНИЙ ПОСТМИТОТИЧЕСКИХ КЛЕТОК | 2014 |
|
RU2725502C2 |
| РНК-НАПРАВЛЯЕМАЯ ИНЖЕНЕРИЯ ГЕНОМА ЧЕЛОВЕКА | 2013 |
|
RU2766685C2 |
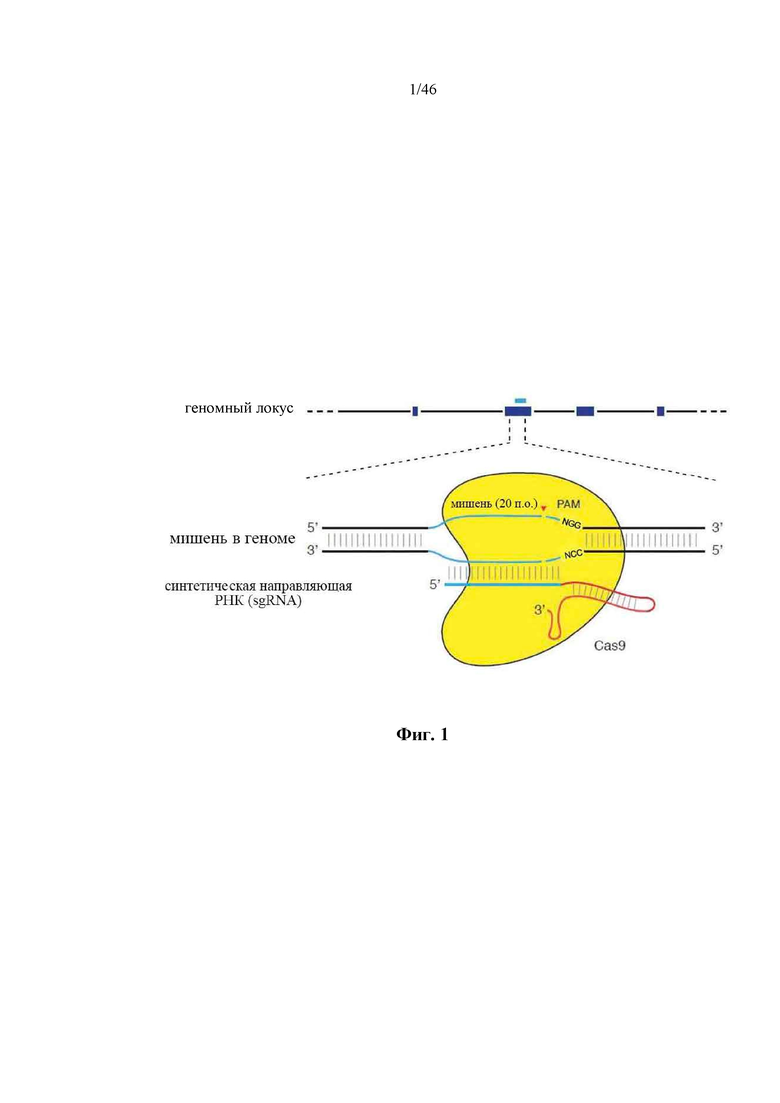
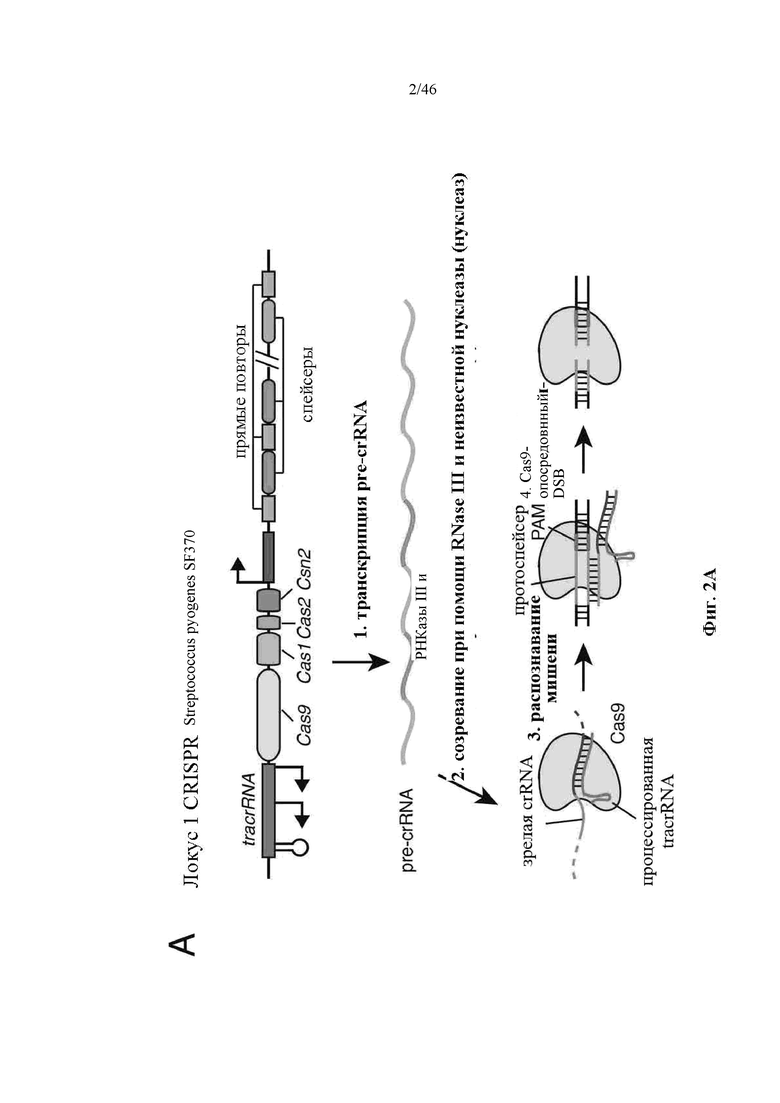
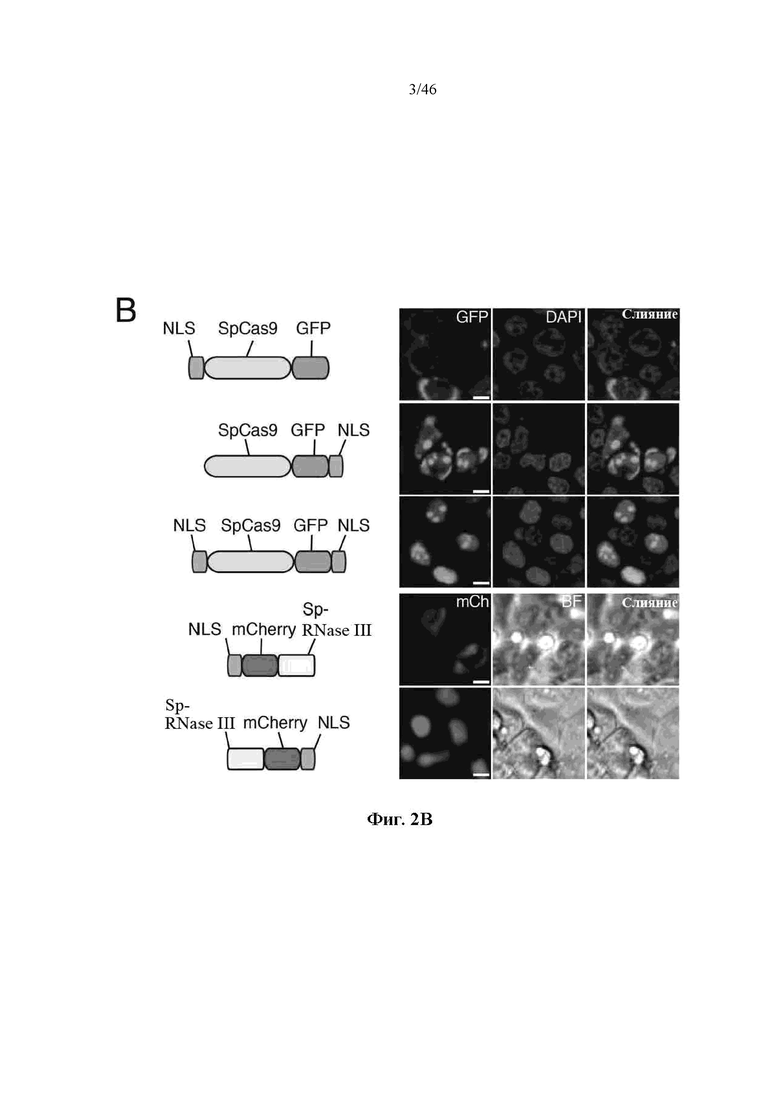

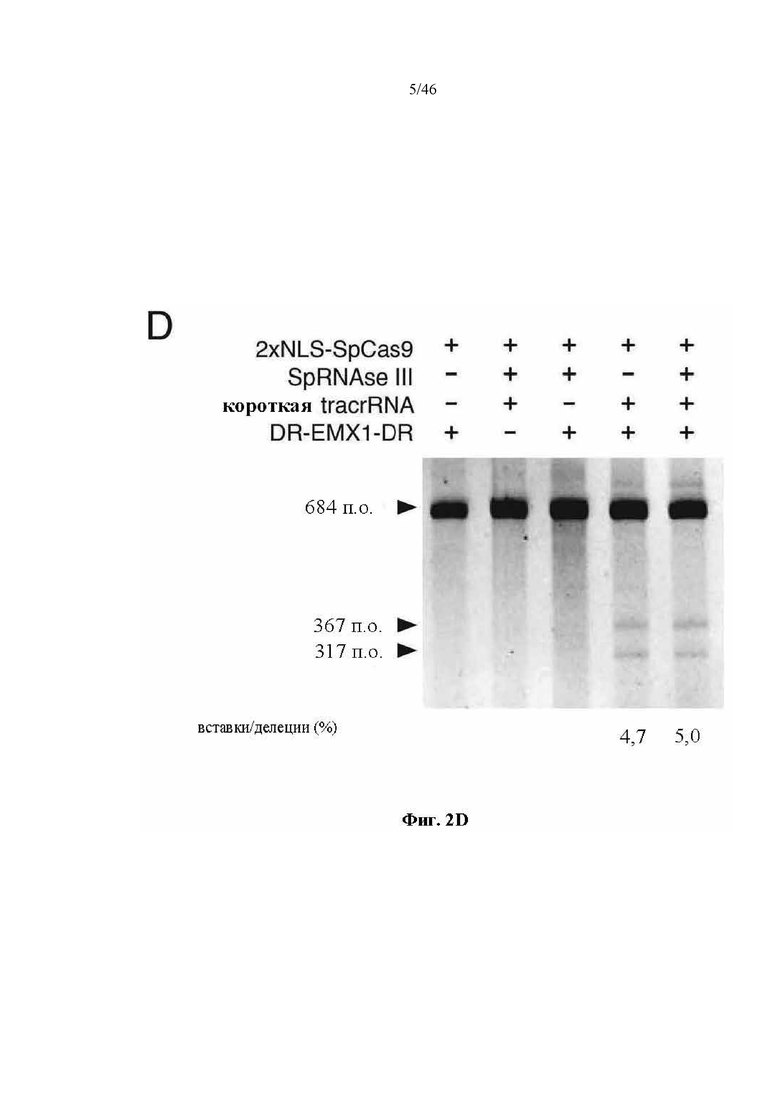
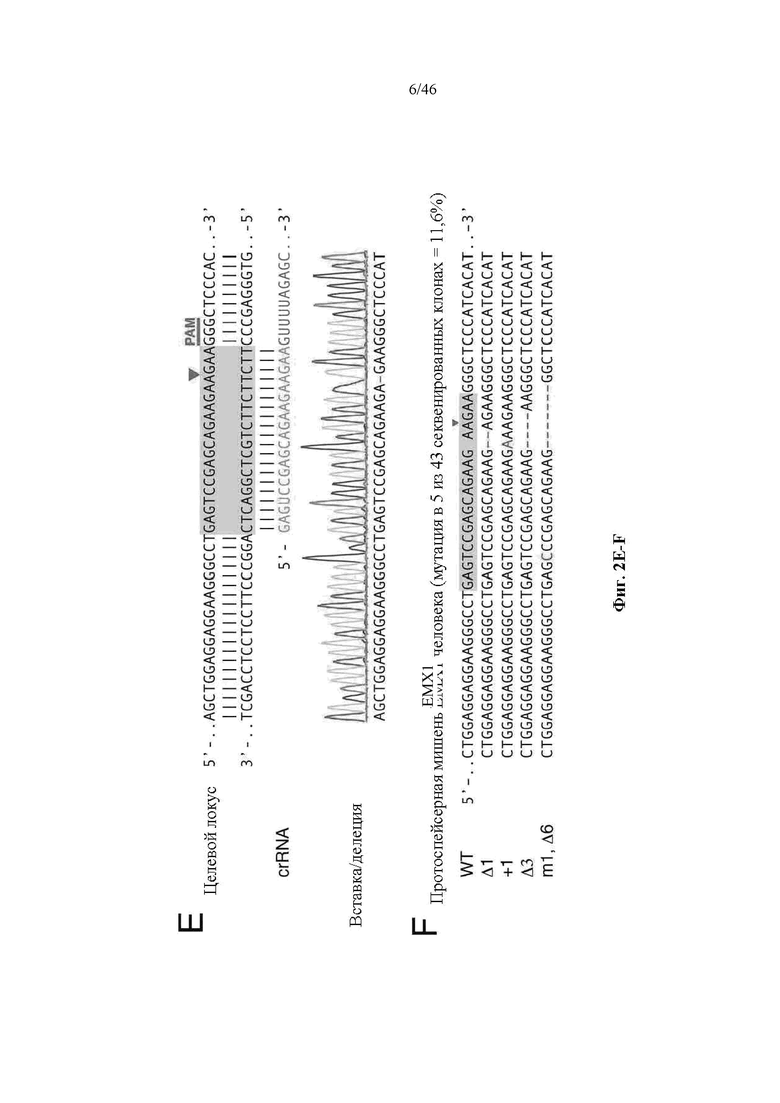
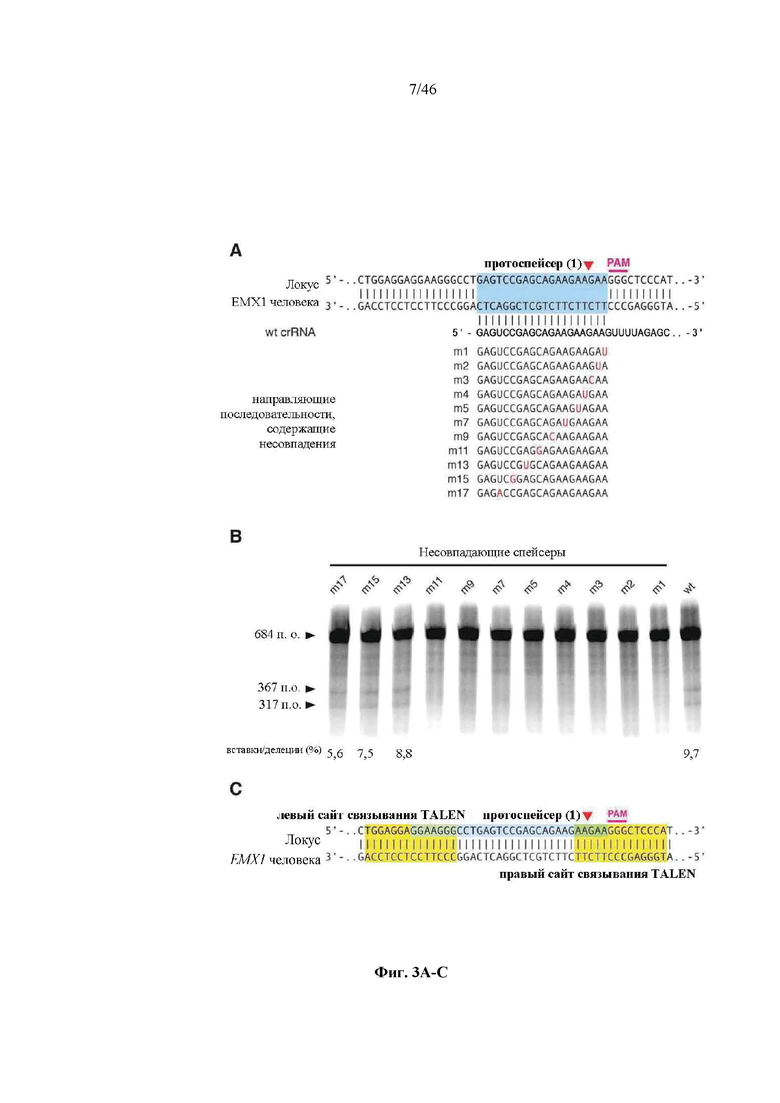
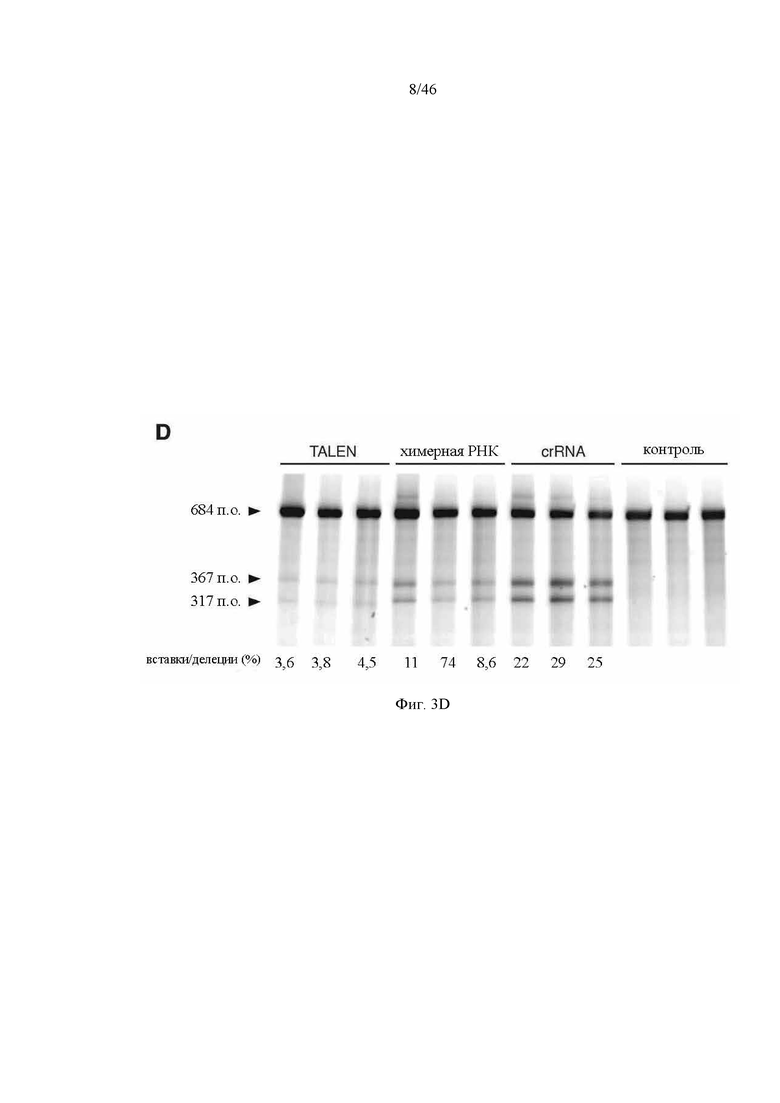
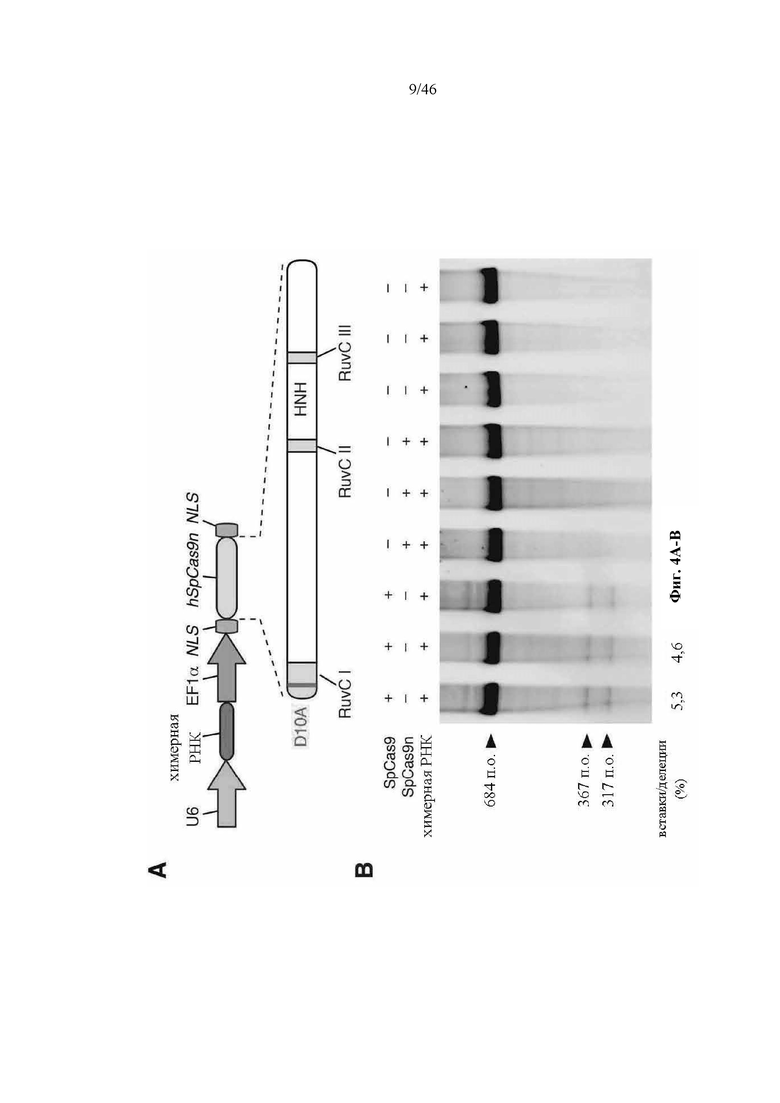
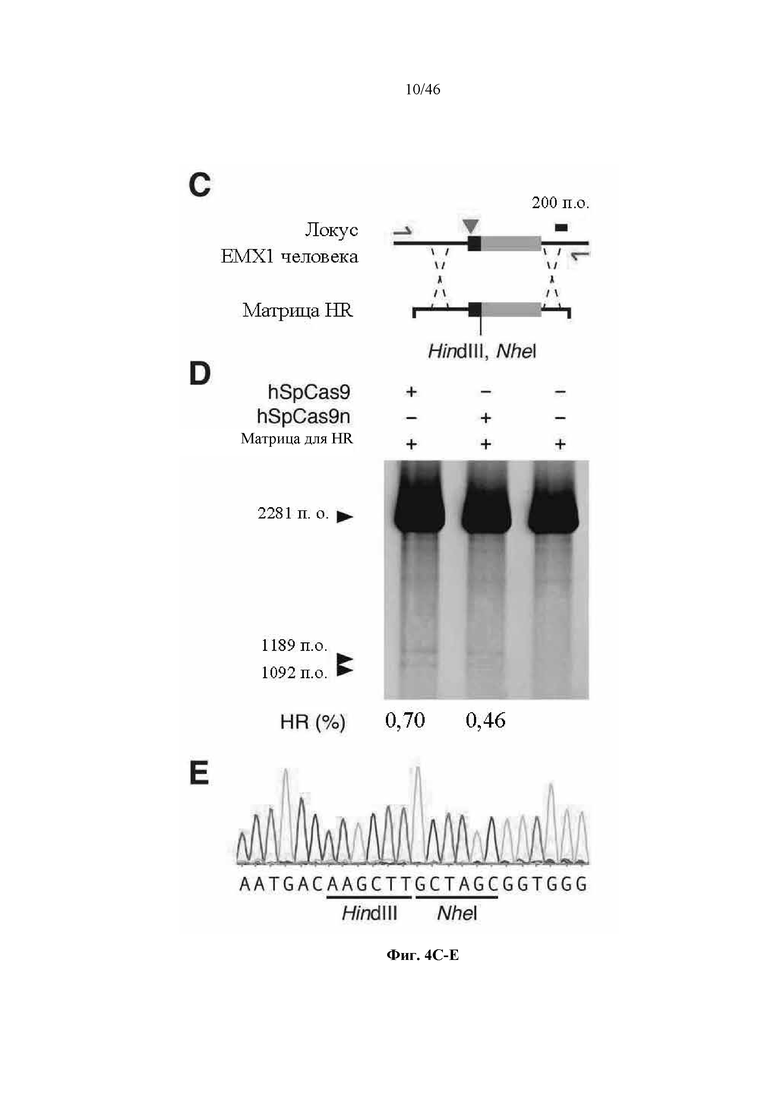
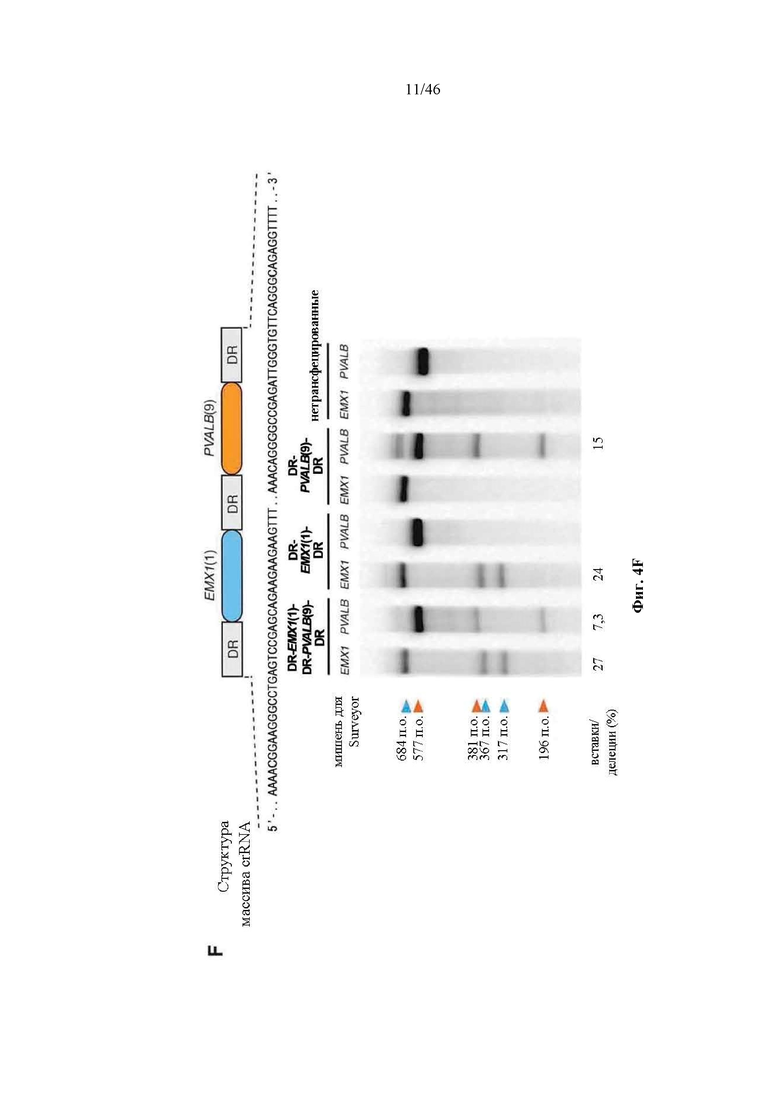
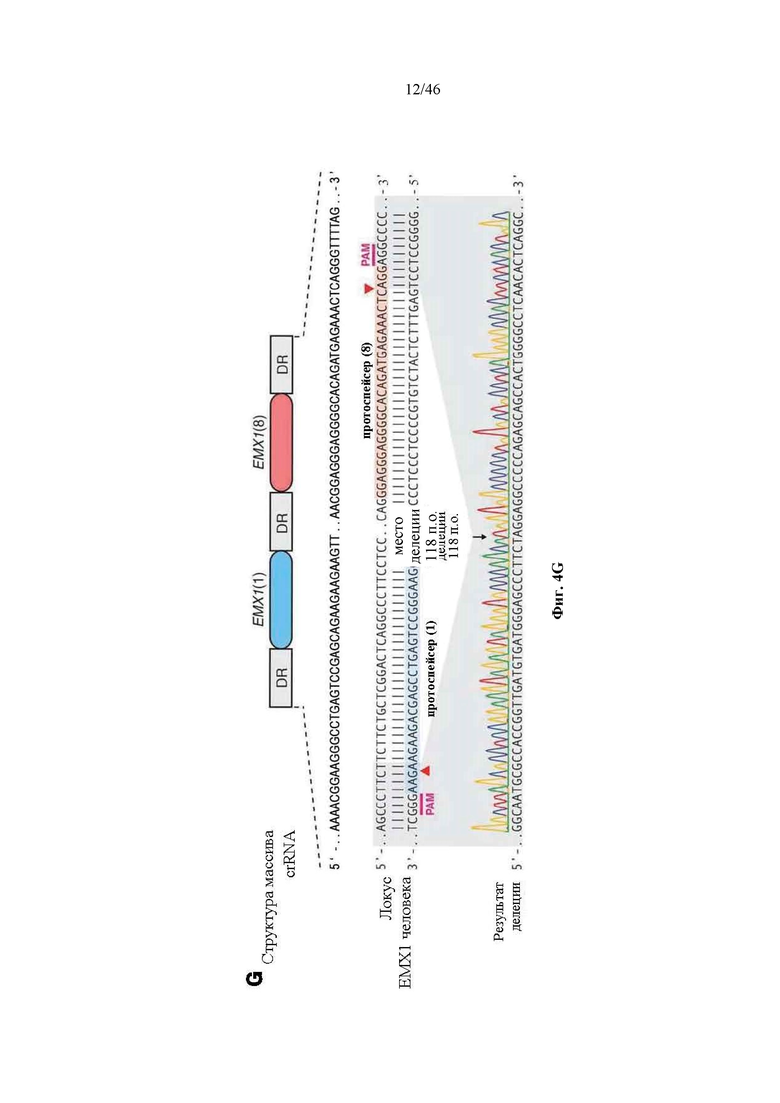
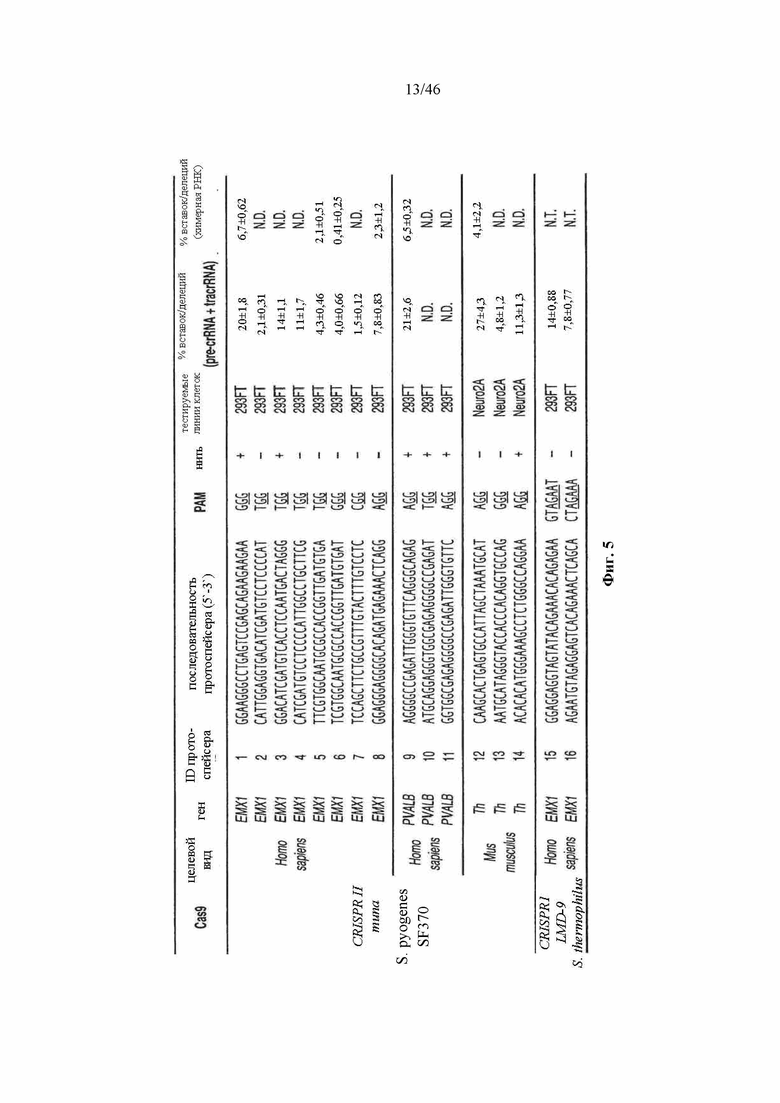
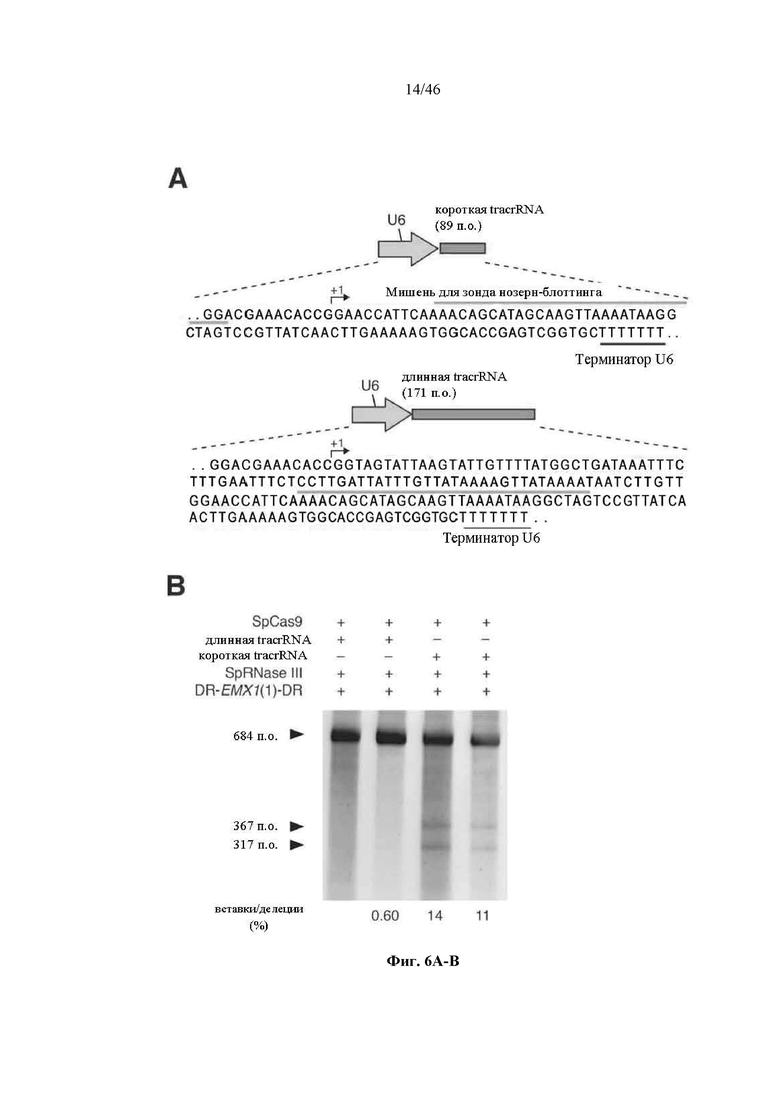
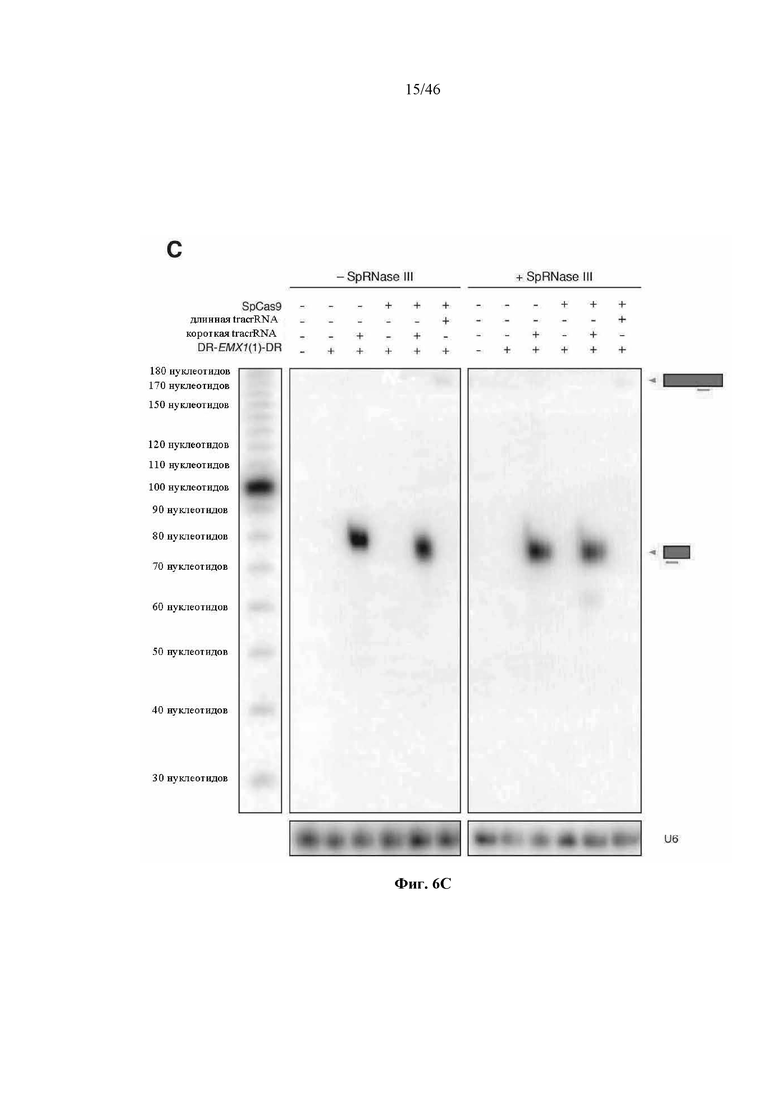

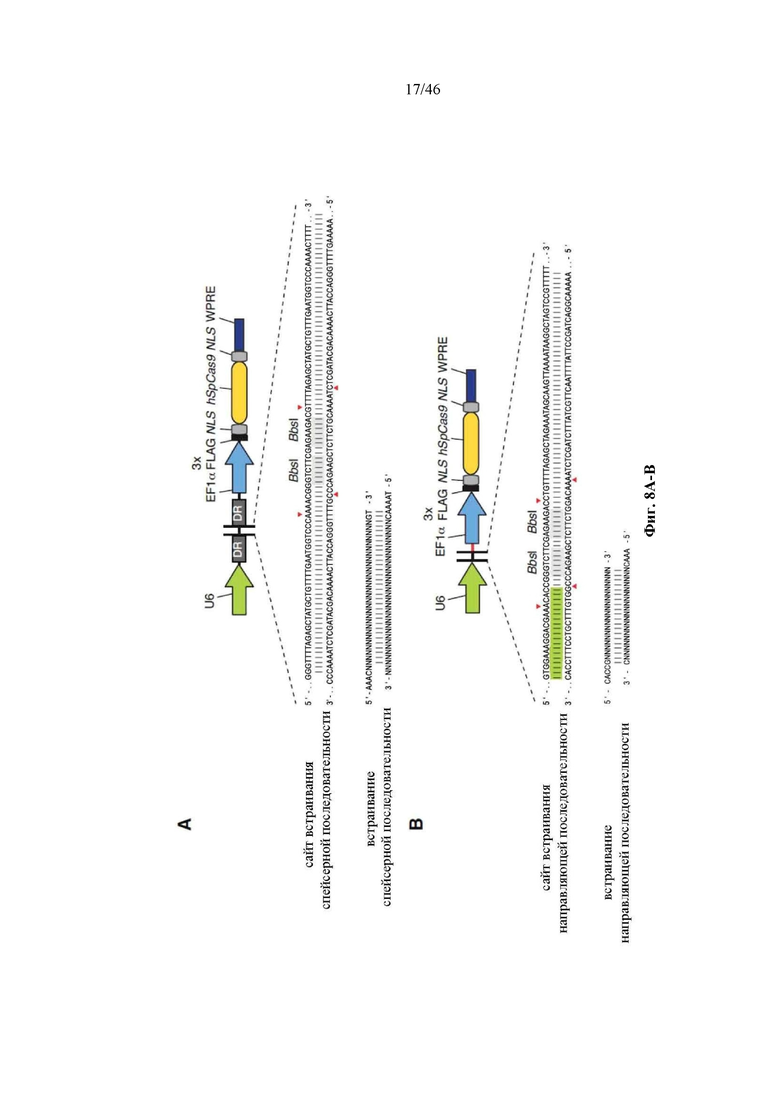
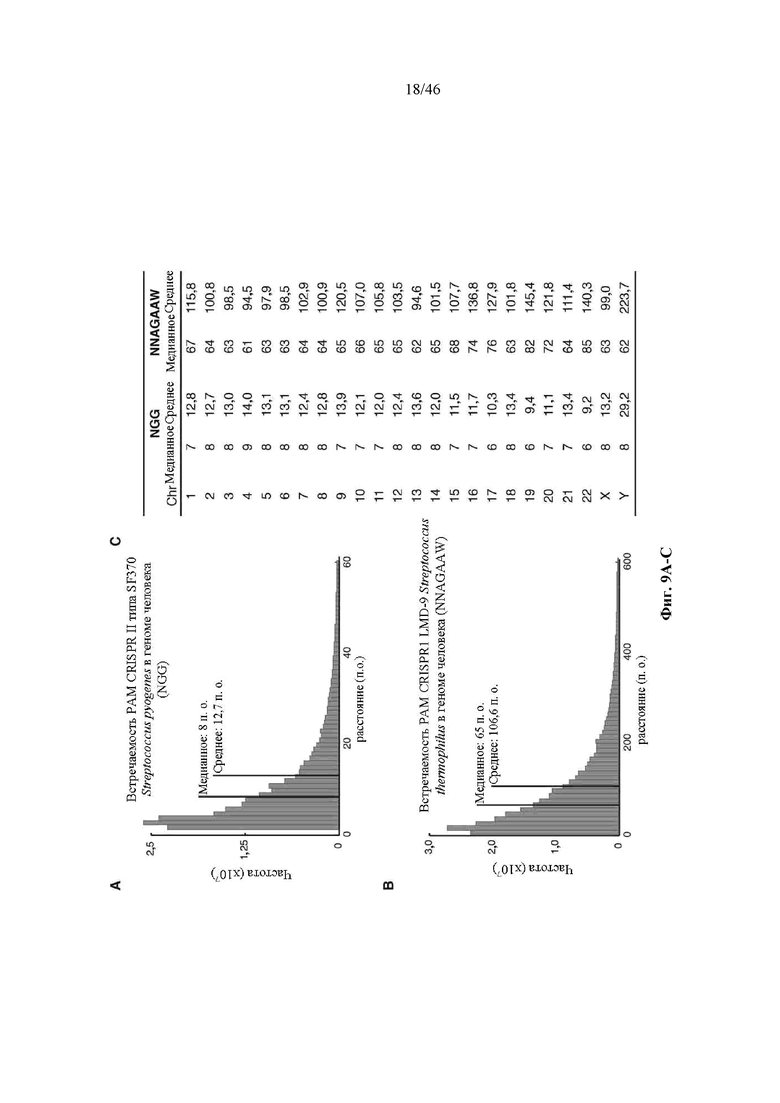
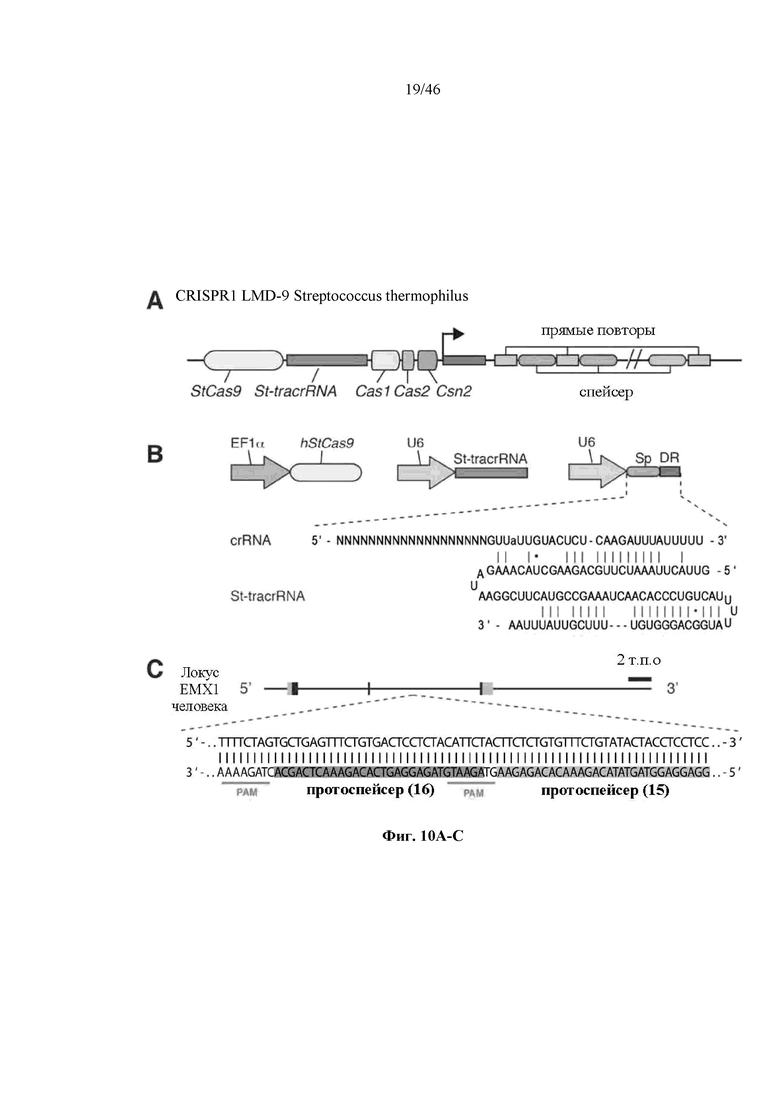
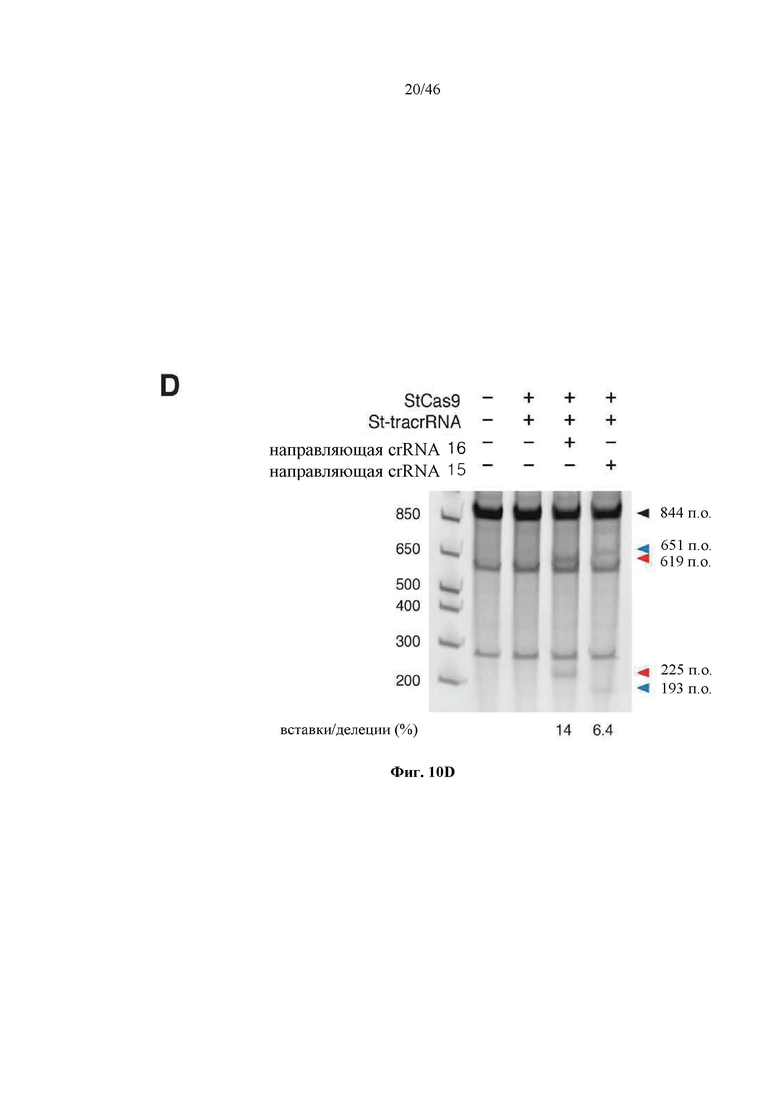

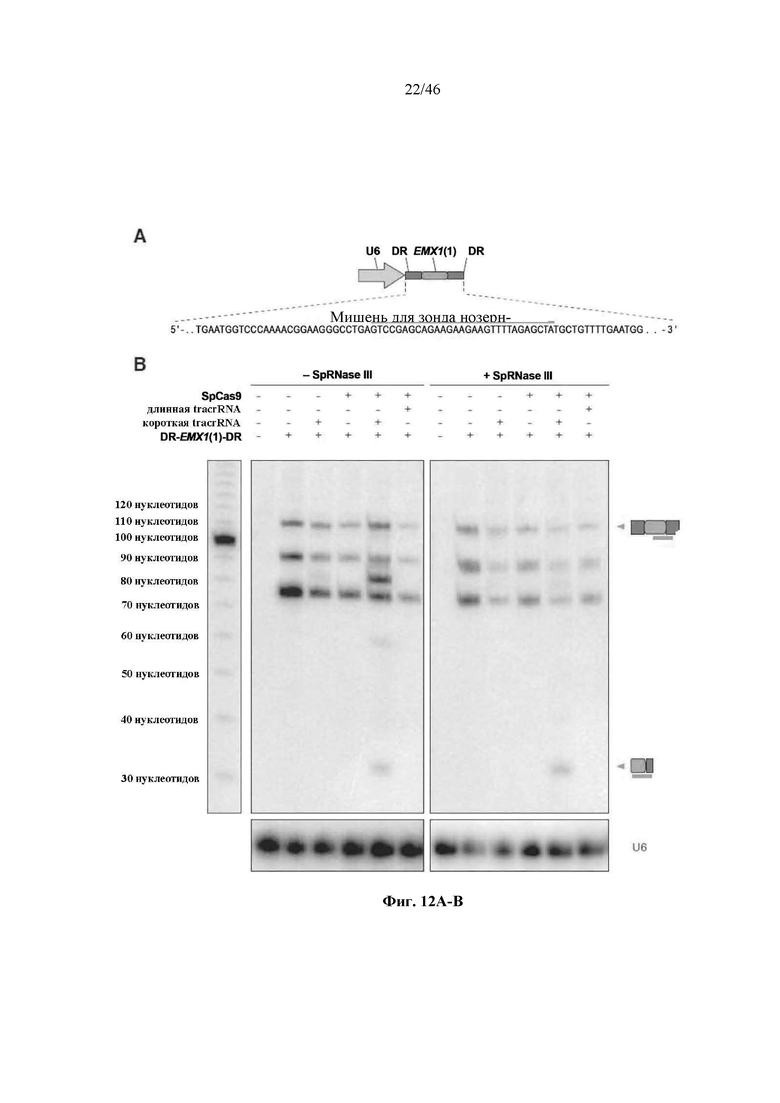
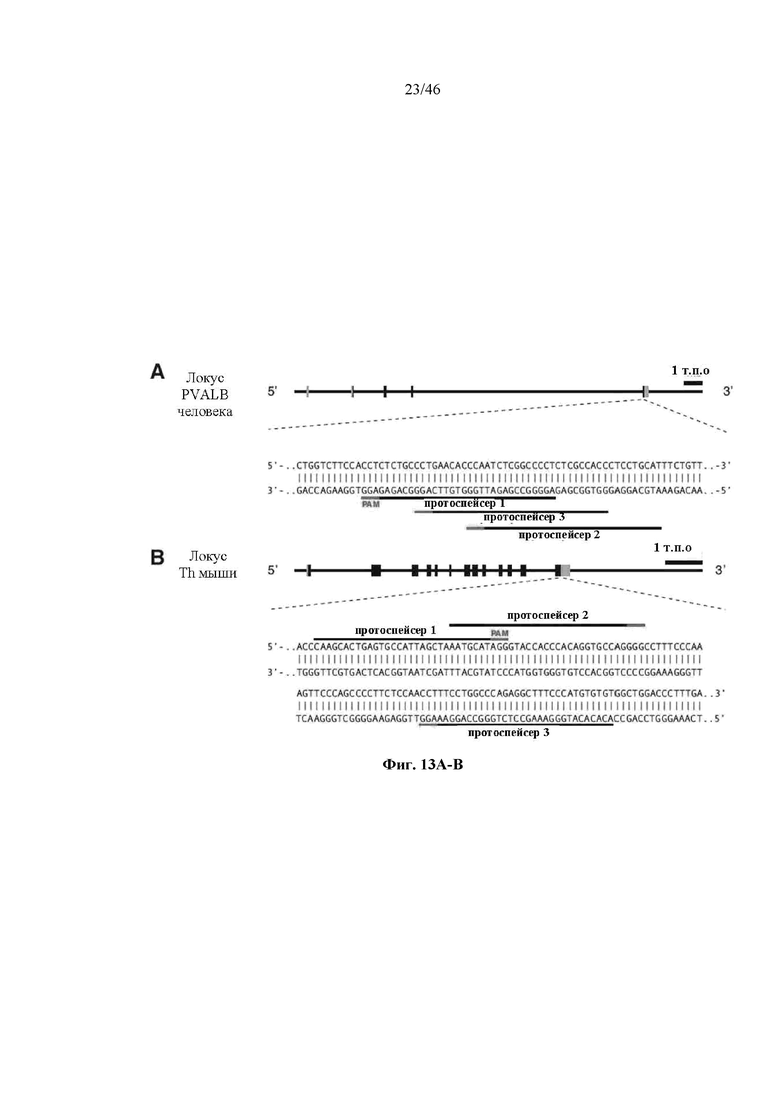

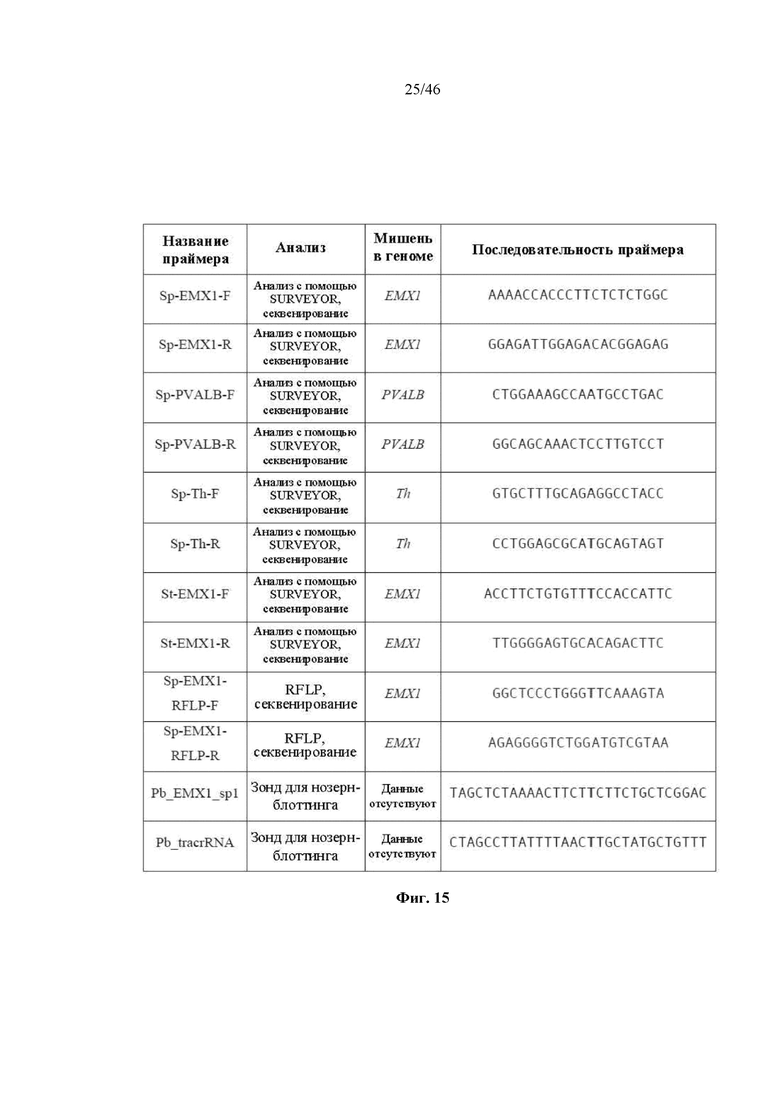
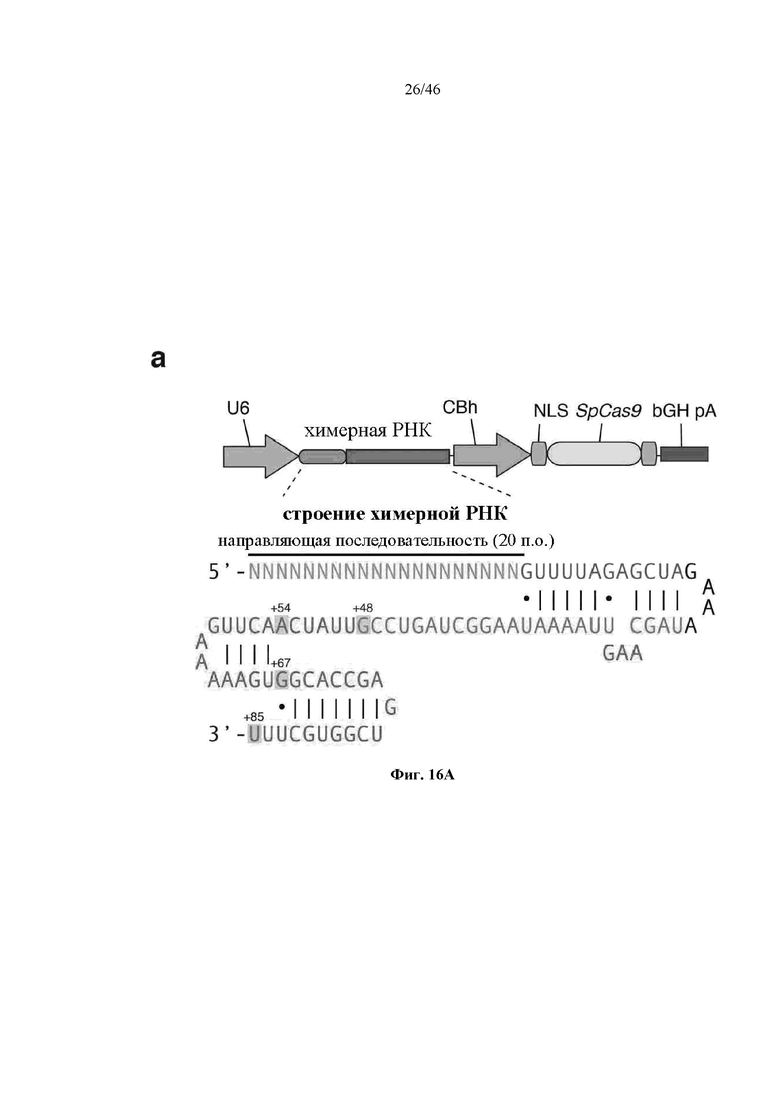
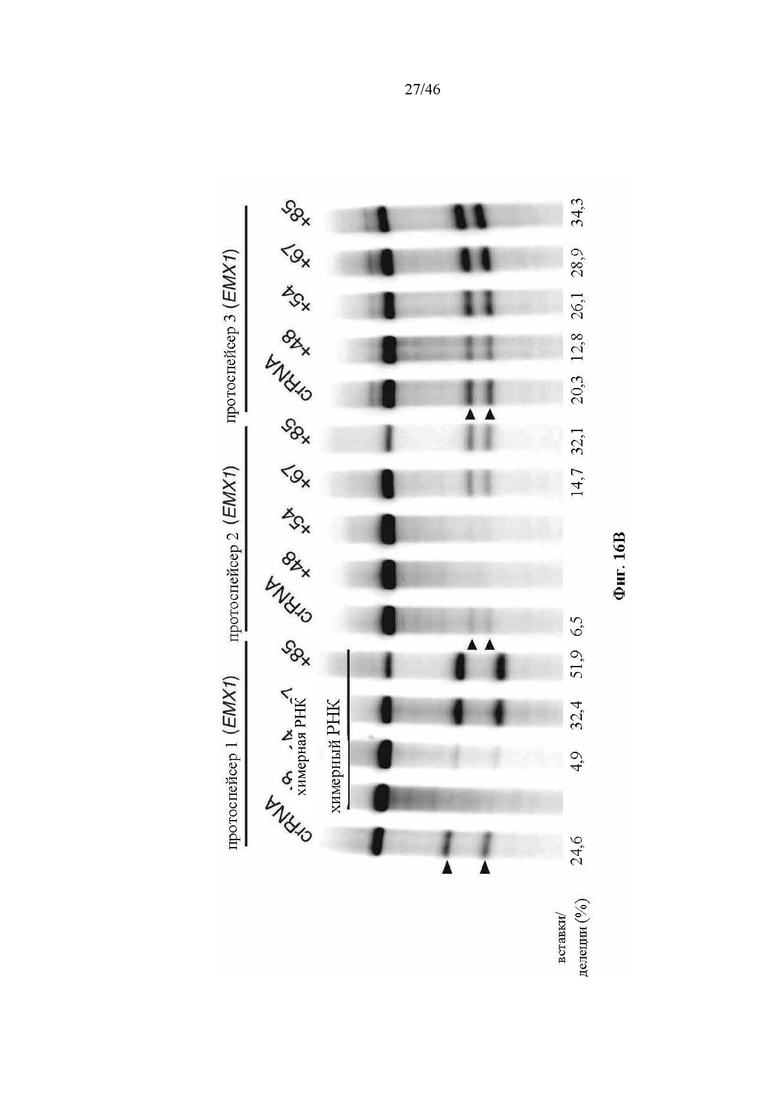
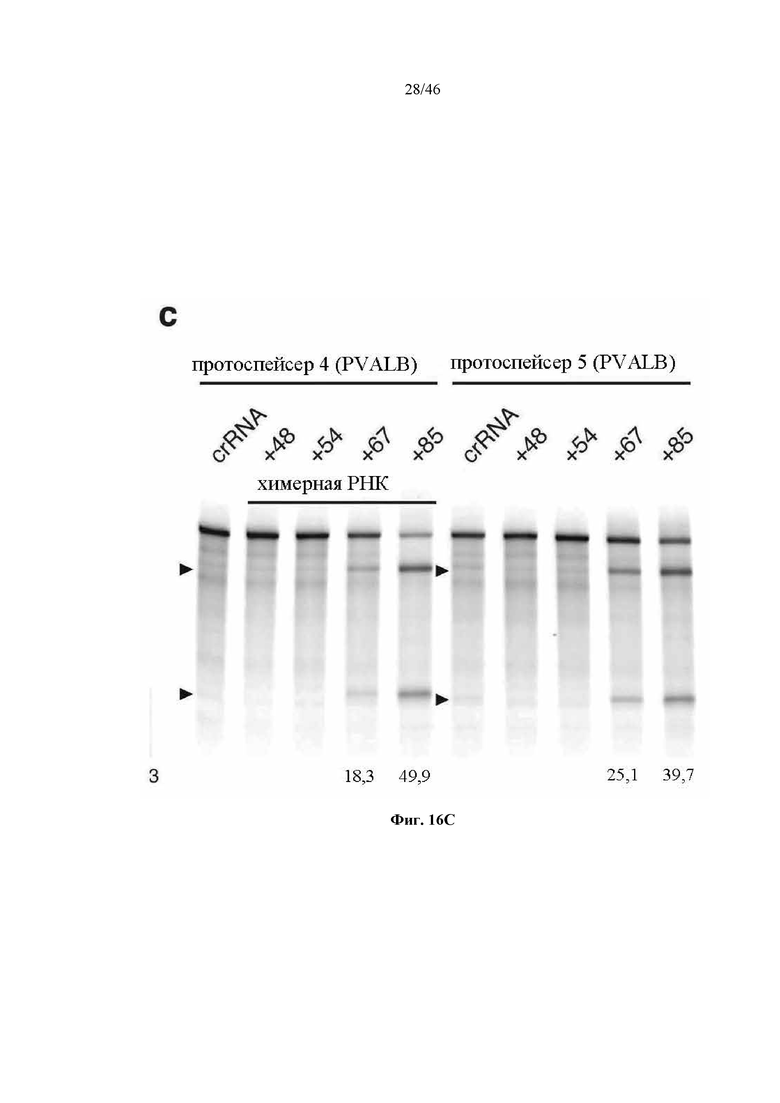
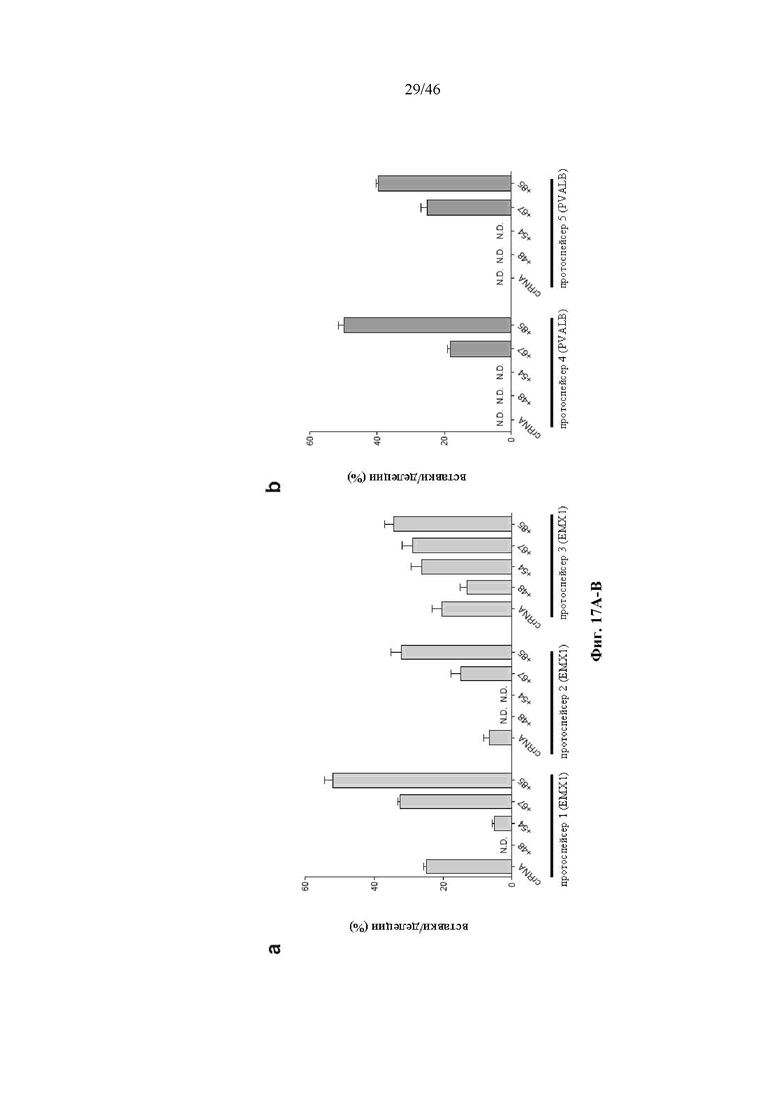

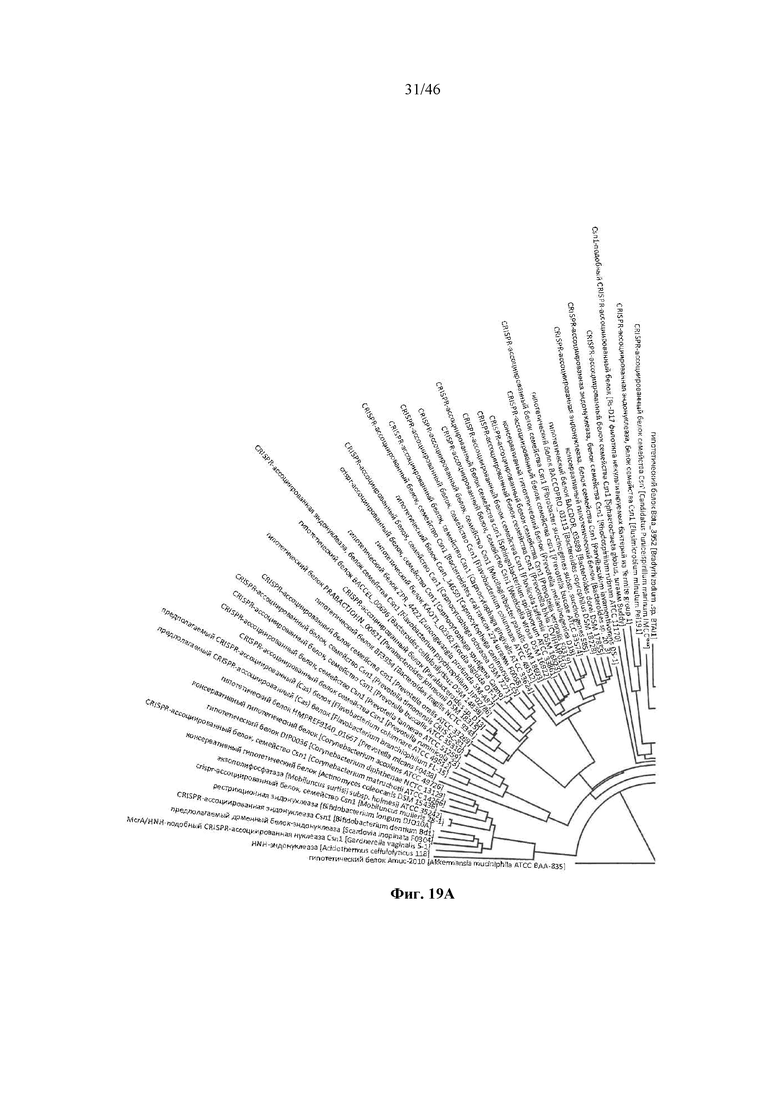

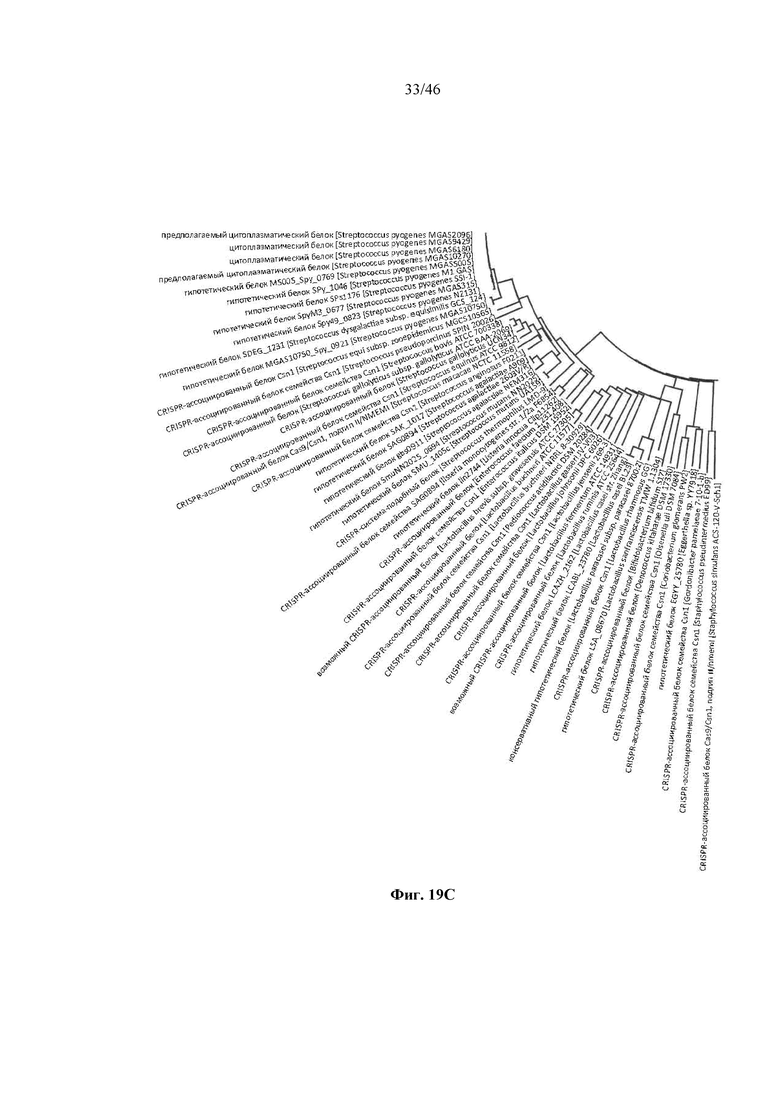







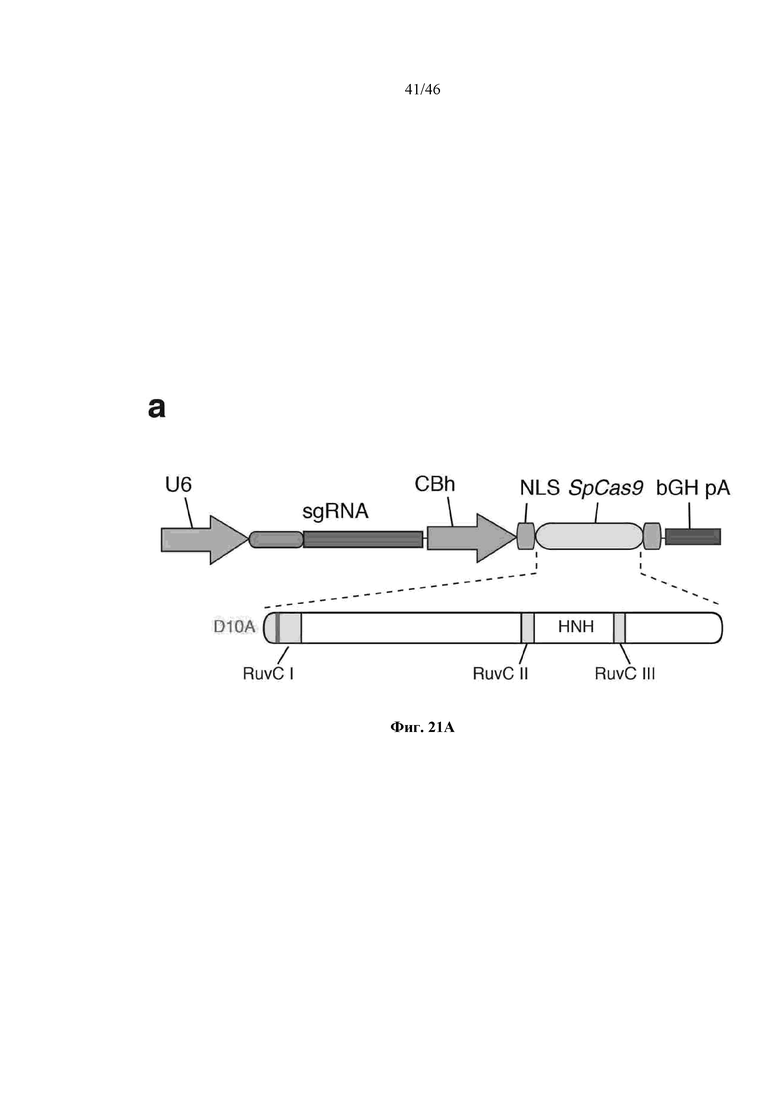
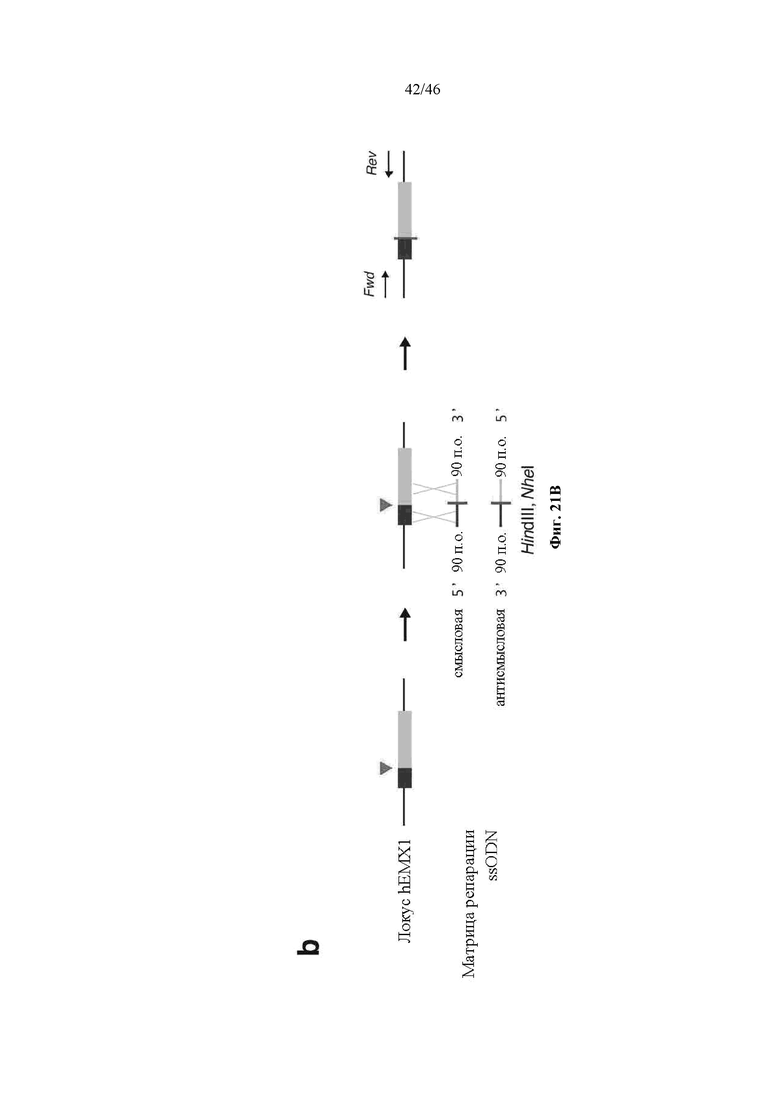
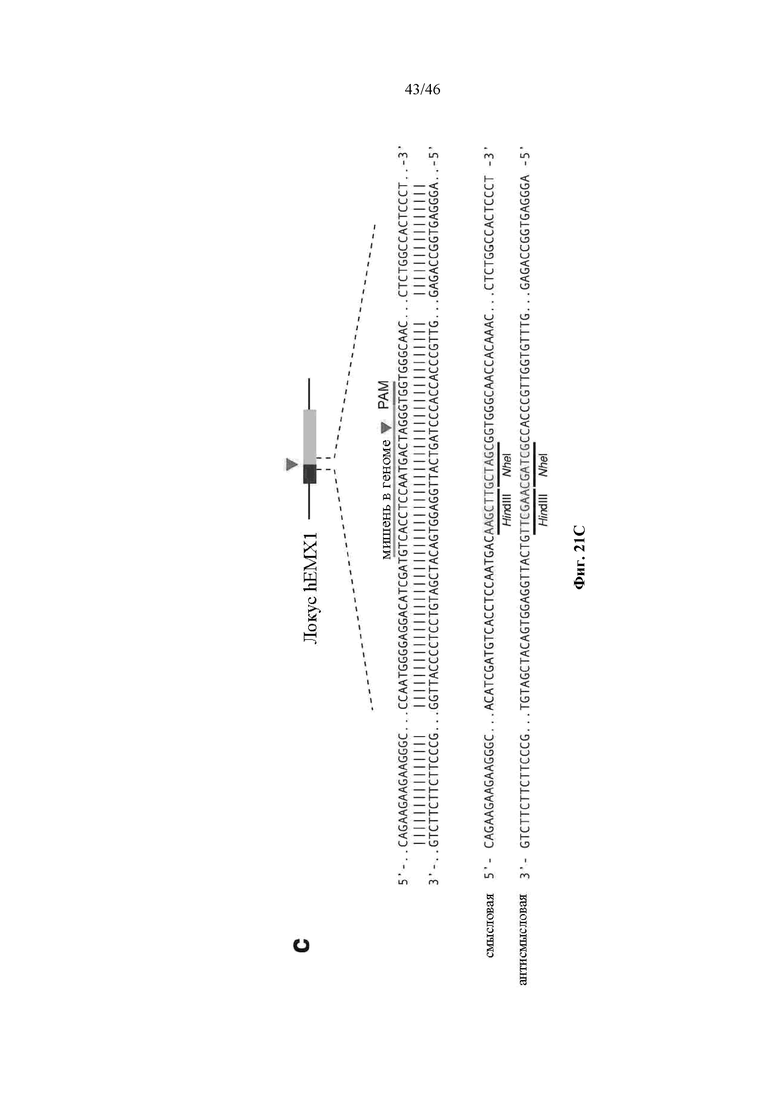
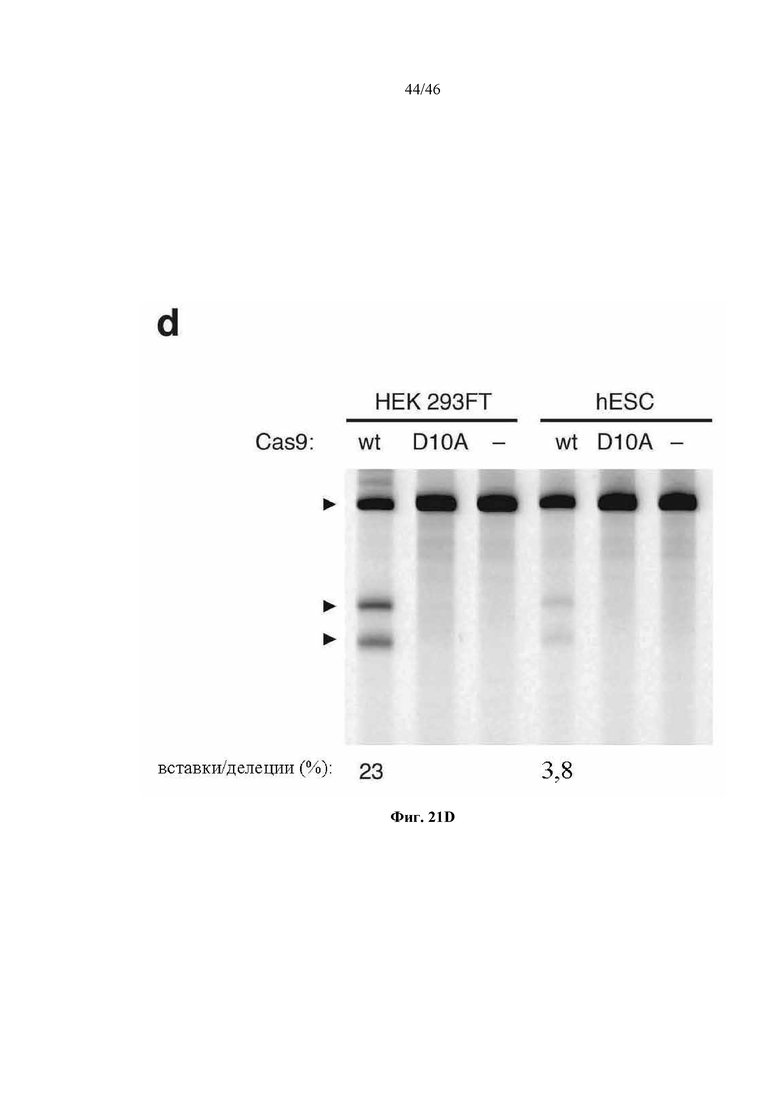
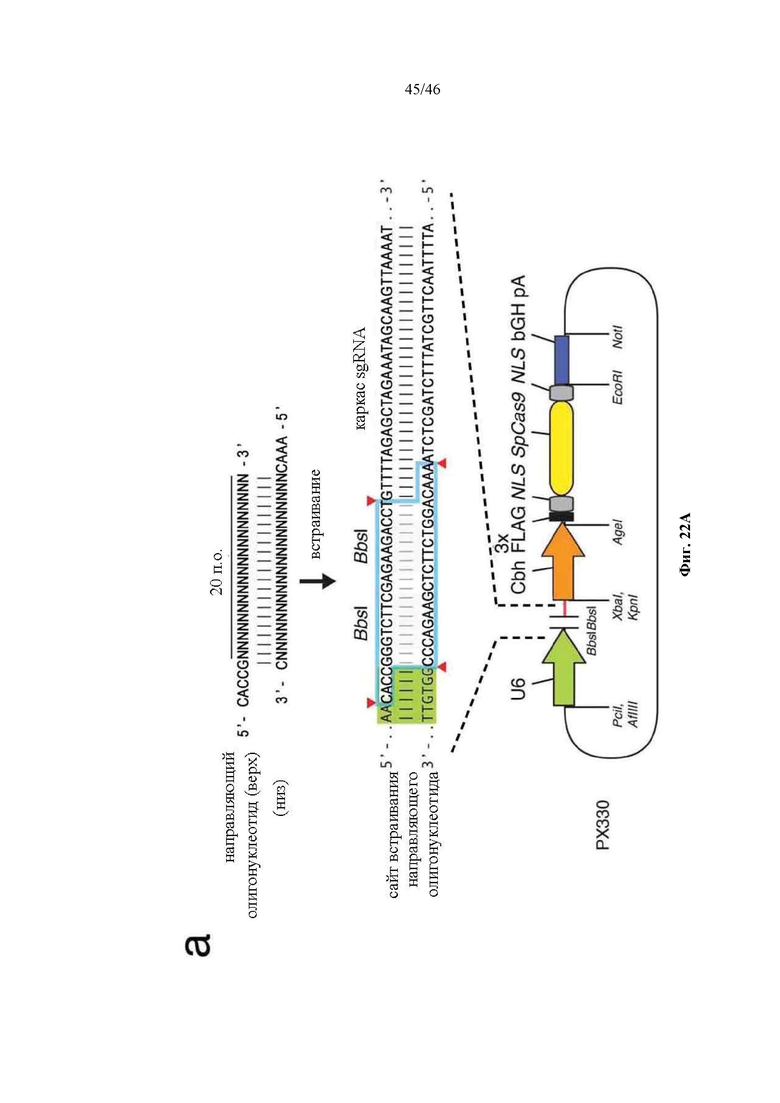
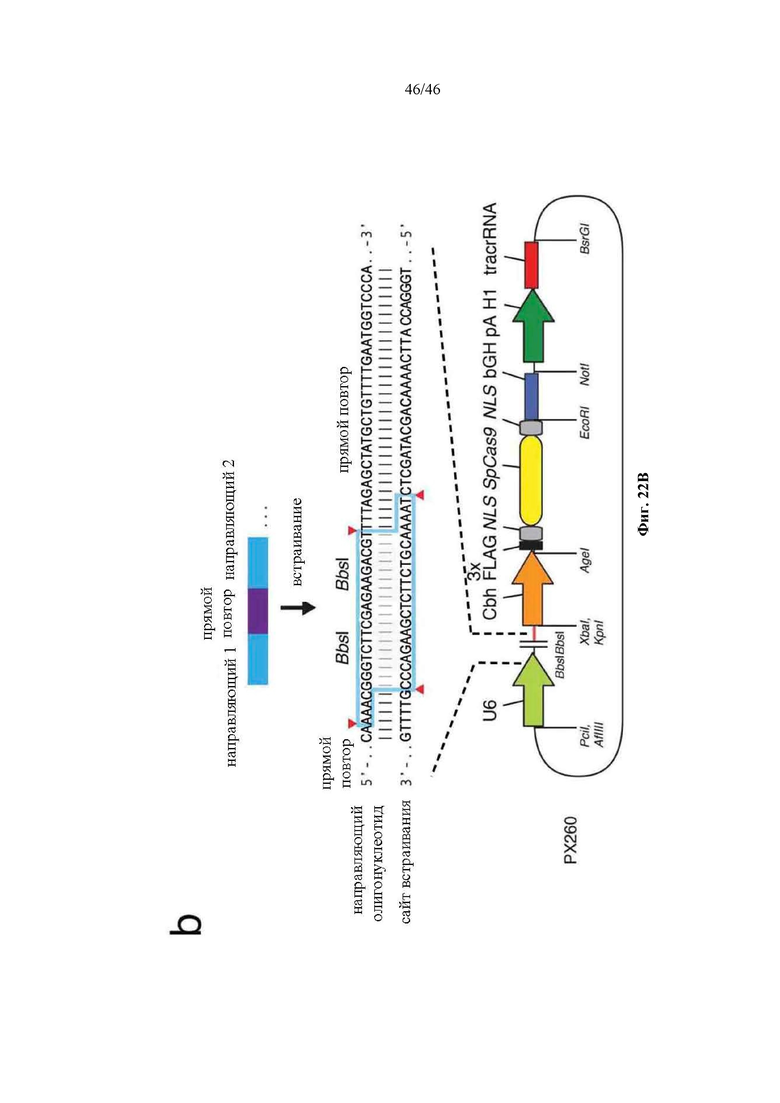
Изобретение относится к области биотехнологии, а именно к сконструированной системе CRISPR-Cas, содержащей белок Cas9 или одну или более молекул нуклеиновых кислот, кодирующих этот белок Cas9, и химерную РНК системы CRISPR-Cas или одну или более молекул нуклеиновых кислот, кодирующих эту химерную РНК. При этом Cas9 слит с по меньшей мере одним сигналом ядерной локализации (NLS), где химерная РНК способна формировать комплекс CRISPR с белком Cas9 и направлять белок Cas9 к целевой последовательности в эукариотической клетке. Изобретение эффективно для модификации эукариотической клетки, которая не является клеткой зародышевой линии человека. 21 з.п. ф-лы, 22 ил., 4 табл., 7 пр.
1. Сконструированная система CRISPR-Cas для модификации эукариотической клетки, которая не является клеткой зародышевой линии человека, содержащая:
(a) белок Cas9 или одну или более молекул нуклеиновых кислот, кодирующих этот белок Cas9, и
(b) химерную РНК системы CRISPR-Cas или одну или более молекул нуклеиновых кислот, кодирующих эту химерную РНК, где указанная химерная РНК содержит в направлении от 5’ к 3’ направляющую последовательность, способную гибридизоваться с целевой последовательностью, примыкающей к мотиву, смежному с протоспейсером, (PAM) в эукариотической клетке, парную tracr-последовательность, которая способна гибридизоваться с tracr-последовательностью, и последовательность tracr, содержащую по меньшей мере 30 нуклеотидов в длину,
где Cas9 слит с по меньшей мере одним сигналом ядерной локализации (NLS), где химерная РНК способна формировать комплекс CRISPR с белком Cas9 и направлять белок Cas9 к целевой последовательности в эукариотической клетке.
2. Система по п.1, отличающаяся тем, что последовательность tracr содержит по меньшей мере 40 нуклеотидов в длину.
3. Система по п.1, отличающаяся тем, что последовательность tracr имеет длину не менее 50 нуклеотидов.
4. Система по любому из пп. 1-3, отличающаяся тем, что Cas9 слит с по меньшей мере двумя NLS.
5. Система по любому из пп.1-4, в которой Cas9 представляет собой Cas9 S. pyogenes, а PAM включает NGG; или где Cas9 представляет собой S. thermophilus Cas9, а PAM включает NNAGAAW.
6. Система по любому из пп.1-5, отличающаяся тем, что Cas9 содержит по меньшей мере одну мутацию в каталитическом домене и представляет собой никазу, не способную расщеплять одну цепь ДНК.
7. Система по п.6, в которой Cas9 содержит по меньшей мере одну мутацию, соответствующую D10A, H840A, N854A или N863A Cas9 S. pyogenes.
8. Система по любому из пп.1-5, отличающаяся тем, что Cas9 содержит по меньшей мере одну мутацию в двух или более каталитических доменах и, по существу, лишен всей активности расщепления ДНК.
9. Система по п.8, в которой Cas9 содержит мутацию, соответствующую D10A Cas9 S. pyogenes, и дополнительно содержит по меньшей мере одну мутацию, соответствующую H840A, N854A или N863A Cas9 S. pyogenes.
10. Система по любому из пп.1-9, в которой Cas9 слит по меньшей мере с одним доменом гетерологичного белка.
11. Система по п.10, отличающаяся тем, что гетерологичный белковый домен содержит эпитопную метку, репортерный домен или белковый домен, обладающий одной или несколькими из следующих активностей: метилазная активность, деметилазная активность, активность активации транскрипции, активность подавления транскрипции, активность фактора высвобождения транскрипции, активность модификации гистонов, активность расщепления РНК и активность связывания нуклеиновых кислот.
12. Система по любому из пп.1-11, в которой химерная РНК содержит один или несколько модифицированных нуклеотидов.
13. Система по любому из пп.1-12, где система содержит химерную РНК и белок Cas9.
14. Система по п.13, в которой белок Cas9 образует комплекс с химерной РНК.
15. Система по любому из пп. 1-12, отличающаяся тем, что указанные одна или более молекул нуклеиновых кислот, кодирующих белок Cas9 представляют собой мРНК.
16. Система по любому из пп.1-12, в которой указанные одна или более молекул нуклеиновых кислот, кодирующих белок Cas9, и указанные одна или более молекул нуклеиновых кислот, кодирующих химерную РНК системы CRISPR-Cas, содержатся в одном или нескольких векторах.
17. Система по п.16, в которой указанные одна или более молекул нуклеиновых кислот, кодирующих белок Cas9, и указанные одна или более молекул нуклеиновых кислот, кодирующих химерную РНК системы CRISPR-Cas, содержатся в одном и том же векторе.
18. Система по п.16 или 17, где векторы представляют собой вирусные векторы.
19. Система по п.18, где вирусные векторы представляют собой ретровирусные, лентивирусные, аденовирусные, аденоассоциированные или векторы вируса простого герпеса.
20. Система по любому из пп. 1-18, отличающаяся тем, что (a) и (b) содержатся в виросоме, липосоме, иммунолипосоме, искусственном вирионе, вирусном векторе или вирусной частице для доставки в эукариотическую клетку.
21. Система по любому из пп.1-20, отличающаяся тем, что целевая последовательность находится в ядре эукариотической клетки.
22. Система по любому из пп. 1-21, отличающаяся тем, что эукариотическая клетка является клеткой млекопитающего или клеткой человека.
| MARTIN JINCK et al., A programmable Dual-RNA-Guided DNA Endonuclease in adaptive bacterial immunity, Science, v.337, n.6096, 17.08.2012, p.816-821 | |||
| GIEDRIUS GASIUNAS et al., Cas9-crRNA ribonucleoprotein comple[ mediates specific DNA cleavage for adaptive immunity in bacteria, Proceedings of the national academy of sciences, v.109, n.39, |
Авторы
Даты
2023-05-22—Публикация
2013-12-12—Подача