Область техники
Изобретение относится к области биотехнологии и генной инженерии, в частности к иммунобиологическому средству на основе вирусоподобных частиц ротавируса А. Средство содержит капсидные гены ротавируса (VP2 и VP6), а также поверхностные белки VP4 и VP7 разных генотипов ротавируса, синтезированные в бакуловирусной системе экспрессии и собранные в трехслойные вирусоподобные частицы (VLP). Предложенное иммунобиологическое средство может применяться для индукции специфического иммунитета против инфекции, вызываемой ротавирусом А человека.
Уровень техники
Актуальность разработки антивирусных препаратов или вакцин для профилактики ротавирусной инфекции не вызывает сомнений. Российская Федерация относится к регионам с высоким уровнем заболеваемости острыми кишечными инфекциями (ОКИ). Среди очагов групповой заболеваемости, вызванных возбудителями с фекально-оральным механизмом передачи, ротавирусная инфекция занимает 2-е место.
Существуют значительные географические различия в распространенности ротавирусов разных генотипов, причем с течением времени происходит изменение встречаемости генотипов на одной и той же территори. Согласно данным референс-центра по мониторингу ОКИ в период с 2012-2020 гг., преобладающими генотипами ротавирусов группы А являются G9P[8], G3P[8], G2P[4], G4P[8], G1P[8], G12P[8].
В настоящее время единственным эффективным методом контроля уровня заболеваемости признается вакцинация [World Health Organization. 2018]. В настоящее время существует три вакцины, прошедшие предварительную квалификацию во Всемирной организации здравоохранения (ВОЗ): моновалентная вакцина (RV1) Rotarix (Glaxo Smith Kline) на базе штамма ротавируса человека, реассортантная пентавалентная (RV5) вакцина RotaTeq (Merck) на основе бычьего ротавируса и моновалентная вакцина Rotavac (Bharat Biotech). С октября 2012 г. в России зарегистрирована вакцина RotaTeq, и входит в календарь профилактических прививок по эпидемическим показаниям РФ [Приказ Минздрава России № 125н от 21.03.2014]. В настоящий момент в некоторых регионах РФ проходит пилотный проект по вакцинации против ротавирусной инфекции [http://24.rospotrebnadzor.ru/press/release/101613/]. Мониторинг циркулирующих штаммов ротавирусов группы А необходим, как для более точного подбора антигенного состава вакцин, так и для оценки возможного воздействия вакцинации на спектр циркулирующих генотипов. Эффективность живых пероральных вакцин оказалась ниже среди малоимущих и странах со средним уровнем дохода в Африке и Азии, чем в странах с высоким доходом, возможно также из-за различия между циркулирующими штаммами и антигенами, входящими в состав вакцин.
В качестве нового направления борьбы против ротавирусной инфекции в последнее время были предложены вакцины на основе рекомбинантных технологий - на основе вирусоподобных частиц или virus like particles (VLP). Вирусоподобные частицы или virus like particle (ВПЧ или VLP) представляют собой структуры размером порядка 100-150 нм, которые являются результатом самосборки вирусных белков без нуклеиновой кислоты генома внутри.
VLP могут вызывать Т-клеточный и В-клеточный иммунный ответы. Из-за большого количества повторяющихся фрагментов вирусных поверхностных и/или коровых белков, представляющих собой конформационные вирусные эпитопы, при иммунизации VLP стимулируют дендритные клетки, которые захватывают соответствующие антигены для презентации T- и В-лимфоцитам [Roy P., Noad R. Virus-like particles as a vaccine delivery system. Myths and facts. Hum. Vaccines. 2008; 4 (1): 5-8.]. Поскольку VLP не могут реплицироваться, они обеспечивают более безопасную альтернативу живым вакцинам на основе аттенуированных вирусов. Вирусные структурные белки в процессе самосборки могут объединяться в организованные макромолекулярные структуры (капсиды), по своему строению и морфологии схожие с природными вирионами и содержащие функционально активные и иммунологически соответствующие структурные белки.
VLP могут продуцироваться в различных экспрессионных системах. Наиболее перспективными считаются VLP на основе белков, полученных в бакуловирусной системе экспрессии. Эта системы дают высокие уровни экспрессии рекомбинантных белков и позволяют организовать последующее крупномасштабное производство вакцины. VLP, продуцируемые в клетках насекомых, считаются более безопасными, чем другие системы экспрессии, поскольку бакуловирусы обнаруживаются в овощах и не способны реплицироваться в клетках млекопитающих. Кроме того, клетки насекомых можно адаптировать для роста в бессывороточной, не содержащей животных продуктах, среде, могут быть идентифицированы с помощью анализа кариотипа и изофермента, свободны от загрязняющих микроорганизмов, случайных агентов, ретровирусов и, как было показано, не являются онкогенными. Считается, что данная технология безопасна и используется для широкомасштабного производства VLP [Rong R, Progress in vaccine development based on baculovirus expression vector system PMID:31001944, DOI:10.13345/j.cjb.180301].
В настоящее время, разработано несколько вакцин, которые находятся на разных стадиях клинических испытаний:
- Парентеральная вакцина Ro-VLP на основе растительных вирусоподобных частиц. [Natsuki Kurokawa et. al. Safety and immunogenicity of a plant-derived rotavirus-like particle vaccine in adults, toddlers and infants// VaccineVolume 39, Issue 39, 2021, PP 5513-5523];
- Трехвалентная парентеральная субъединичная ротавирусная вакцина VP2-P8 [Michelle J Groome et. al. Safety and immunogenicity of a parenteral trivalent P2-VP8 subunit rotavirus vaccine: a multisite, randomised, double-blind, placebo-controlled trial// Lancet Infect Dis 2020; 20: 851-63].
Эти вакцины охватывают единичные генотипы ротавируса, что недостаточно для эффективной профилактики, как минимум 6-ти генотипов ротавируса, циркулирующих в РФ.
Кроме того, что касается субъединичной вакцины, то недостатком рекомбинантных субъединичных вакцин, является то, что презентация антигена может отличаться от природного вируса и будут вырабатываться антитела, не способные эффективно нейтрализовать патоген.
Наиболее близким аналогом заявленного изобретения является документ (EP 1261629 B1, опуб. 02.11.2006), в котором раскрывается техническое решение, относящиеся к рекомбинантному бакуловирусу, содержащему, экспрессирующие белки VP2, VP6, VP7 и/или VP4 ротавируса, а также содержащему капсидный ген MS2. Также в материалах патента раскрыто получение вирусоподобных частиц на основе бакуловируса. Недостатком данного технического решения является то, что данная конструкция содержит поверхностные антигены ротавируса (VP7 и/или VP4), и Р6 и Р2 одного генотипа, следовательно, не вызывает образование антител против поверхностных белков ротавируса, специфичных для России генотипов ротавируса, позволяющий сформировать специфический иммунный ответ.
Таким образом, существует потребность в создании VLP, сформированных нуклеокапсидом ротавируса (белки VP2, VP6) и поверхностными белками ротавируса 6 генотипов VP4 и VP7 (G1, G2, G4, G9, P4, P8), наиболее представленных на территории России позволяющими формировать 6 типов VLP, которые смогут эффективно формировать широкий спектр антител, и, следовательно, вызывать специфический иммунитет к вирусным генотипам, циркулирующим в Российской Федерации.
Раскрытие сущности изобретения
Технической задачей изобретения является разработка иммунобиологического средства, содержащего VLP, сформированных нуклеокапсидом ротавируса (белки VP2, VP6) и поверхностными белками ротавируса 6 генотипов VP4 и VP7 (G1, G2, G4, G9, P4, P8), и обладающего возможностью эффективно формировать специфический иммунитет к наиболее представленным на территории России 6-ти генотипов ротавируса А.
Технический результат заявленного изобретения заключается в расширении арсенала иммунобиологических средств для индукции специфического иммунитета против инфекции, вызываемой ротавирусом А человека и заключается в создании средства, обладающего высокой степенью безопасности (как белковый препарат) и специфической иммуногенностью и эффективностью для профилактики ротавирусной инфекции, вызываемой определенными генотипами ротавируса А.
Указанный технический результат достигается за счет того, что создано иммунобиологическое средство, содержащее адъювант и несколько рекомбинантных вирусоподобных трехслойных частиц, состоящих из двух белков капсидных генов ротавируса VP2 и VP6, имеющие нуклеотидные последовательности SEQ ID NO:1, SEQ ID NO:2, и одного или несколько из поверхностных белков VP4 и VP7 различных генотипов ротавируса G1, G2, G4, G9, P4, P8, имеющие нуклеотидные последовательности SEQ ID NO:3, SEQ ID NO:4, SEQ ID NO:5, SEQ ID NO:6, SEQ ID NO:7, SEQ ID NO:8, которые получены в бакуловирусной системе экспрессии, при этом вирусоподобная частица имитирует вирион ротавируса А.
В частном случаи исполнения адъювант может представлять собой масляный адъювант, водно-масляную эмульсию, в частности водно-масляную эмульсию на основе сквалена.
Краткое описание чертежей
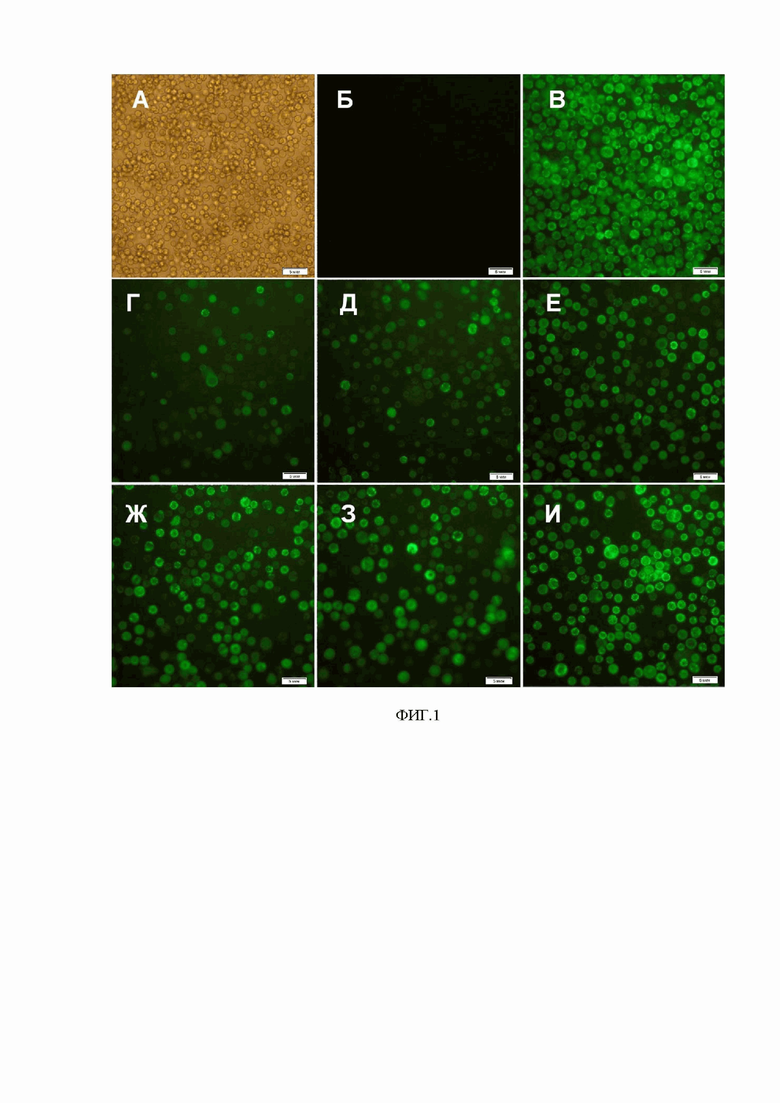
На фиг. 1 представлены результаты заражения бакуловирусом eGFP-Delta92-VP2 суспензионной культуры клеток насекомых T.ni (А. Культура клеток T.ni, в проходящем свете. Б. Культура клеток T.ni, зараженных бакуловирусом дикого типа, в флуоресцентном поле. В. Культура клеток T.ni, зараженных бакуловирусом eGFP-Delta92-VP2/VP6, в флуоресцентном поле). Результаты заражения культуры клеток насекомых T.ni рекомбинантными бакуловирусами с различными вставками в одинаковых дозах (Г-Е) и в оптимизированных дозах (Ж-И): Г,Ж - VP2/VP6+VP7(G1)+VP4(P8); Д, З - VP2/VP6+VP7; Е,И - VP2/VP6+VP4.
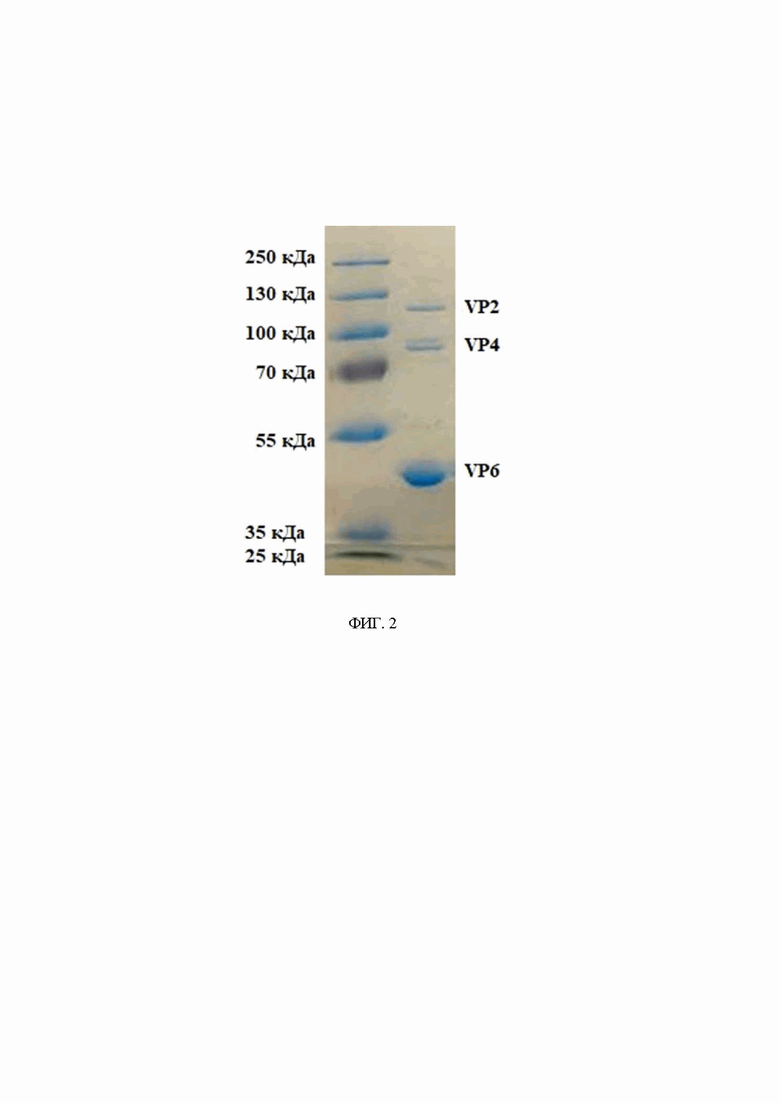
На фиг. 2 показан пример определения чистоты препарата VLP методом ЭФ в ПААГ. На электрофореграмме видны 3 мажорных полосы, соответствующих по молекулярной массе белкам ротавируса А.
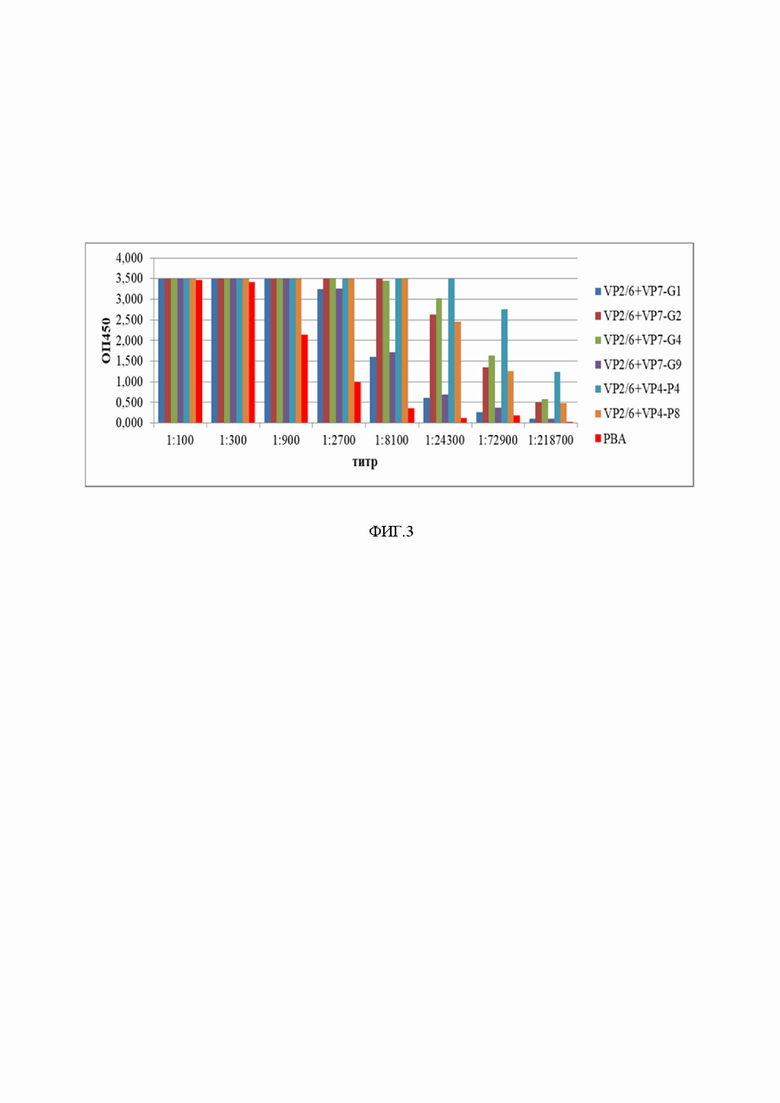
На фиг. 3 представлены результаты измерения специфической активности очищенных VLP и РВА методом ИФА
На фиг. 4 представлены результаты визуализации вирусоподобных и вирусных частиц методом электронной микроскопии (указаны бакуловирусы, которыми были коинфицированы клетки T. ni.) А. VP2/VP6 Б. VP2/VP6+VP4(P8) В. VP2/VP6+VP4(P8) Г. VP2/VP6+VP7(G1) Д. VP2/VP6+VP7(G2) Е. VP2/VP6+VP7(G4) Ж. VP2/VP6+VP7(G9) З. VP2/VP6+VP4(P8)+VP7(G1) И. Ротавирус A
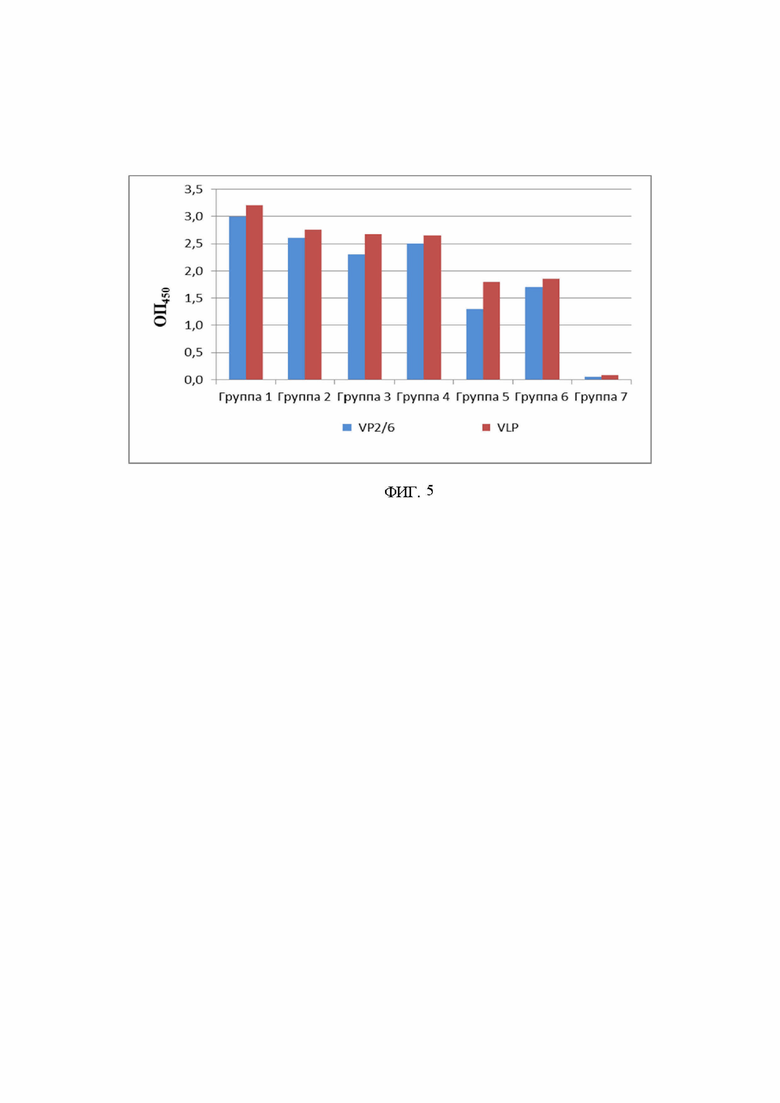
На фиг. 5 описаны результаты обнаружения специфических IgG к белку VP6 и VLP РВА при различном способе введения с адъювантом и без адъюванта методом ИФА
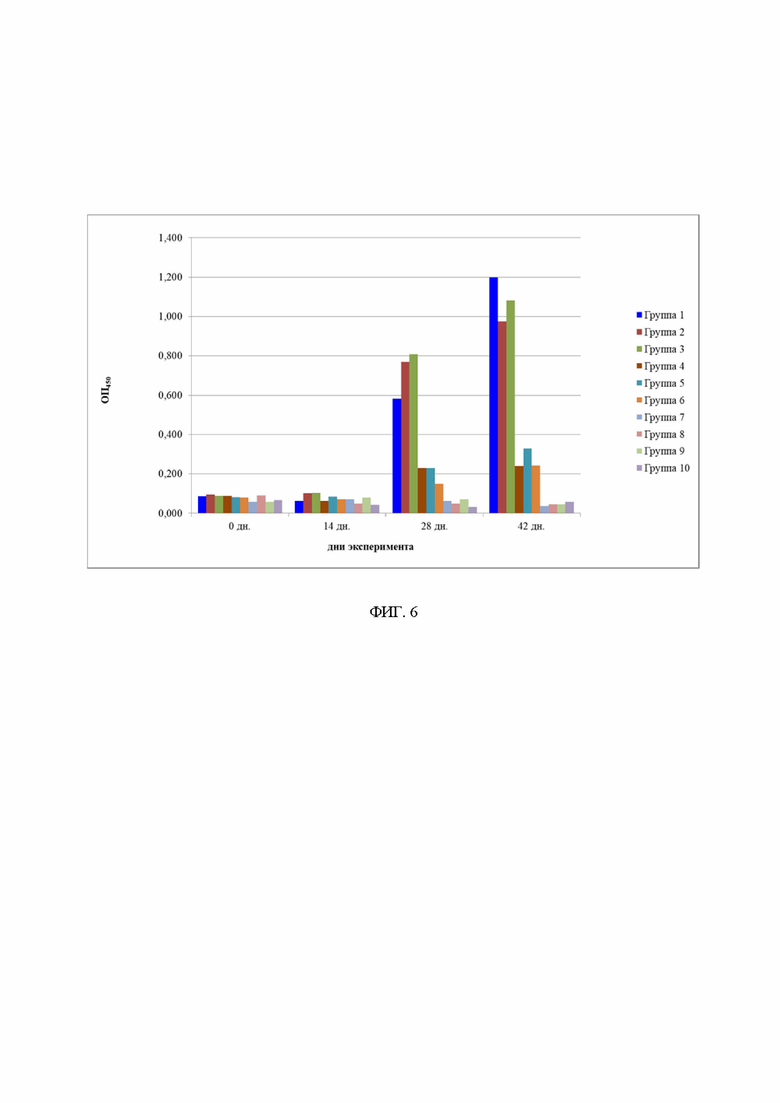
На фиг. 6 показано выявление специфических IgG к белку VP6 РВА методом непрямого ИФА в сыворотках крови поросят на 0, 14, 28 и 42 день эксперимента. Представлены графики средних значений по каждой группе.
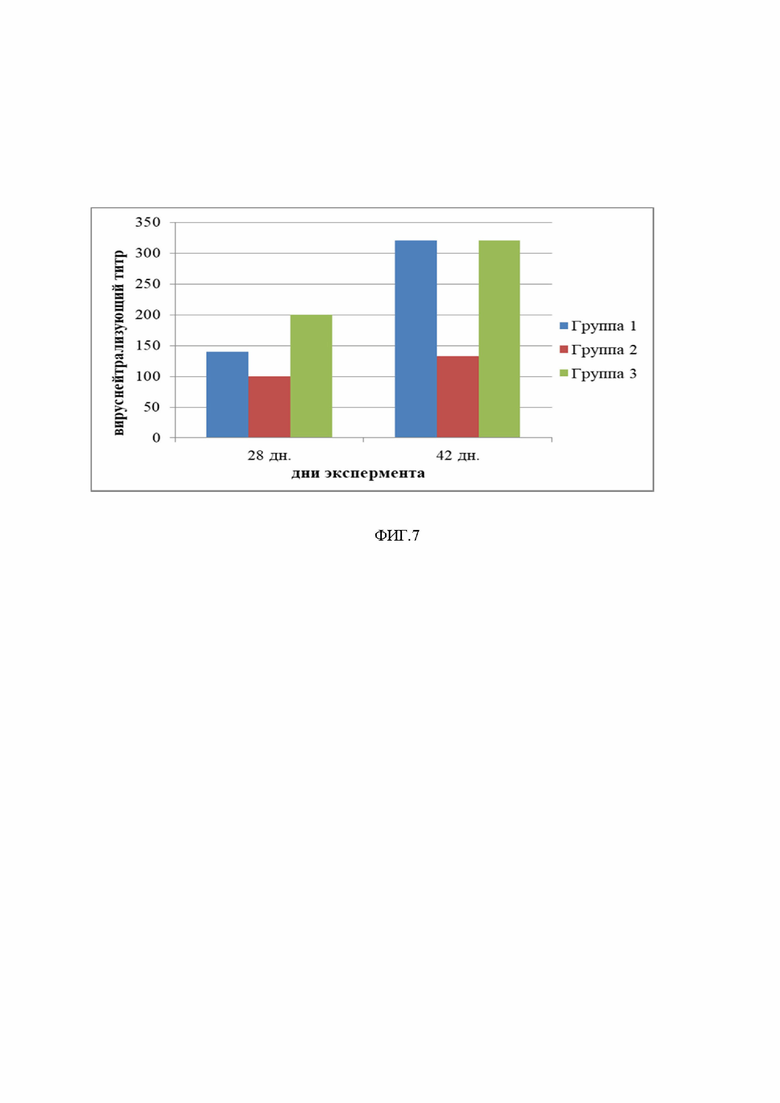
На фиг. 7 представлены результаты измерения титра вируснейтрализующих антител в РН сывороток поросят на 28 и 42 день иммунизации для групп 1-3. - внутримышечное введение VLP (концентрации 30, 150 и 750 мкг/доза, соответственно)
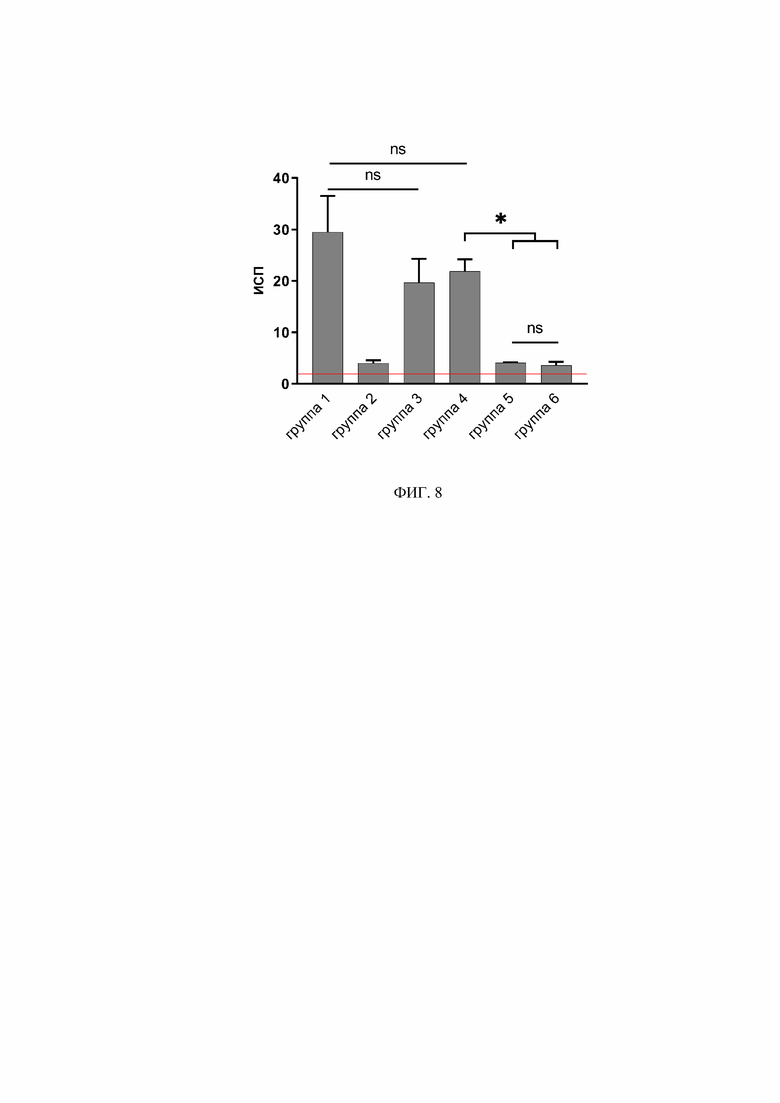
На фиг. 8 представлены результаты реакции бласттрансформации моноцитов периферической крови поросят после первой иммунизации. По оси абсцисс указаны группы животных: 1-3 группы - внутримышечное введение VLP (концентрации 30, 150 и 750 мкг/доза, соответственно), 4-6 группы интраназальное введение VLP в комбинации с атт. штаммом (концентрации 30, 150 и 750 мкг/доза, мкг/доза, соответственно). По оси ординат приведены значения ИСП. *- статистически значимые различия p <0,05, ns - значения ИСП не различаются статистически значимо p >0,05.
Осуществление изобретения
Ранее нами было изучено генетическое разнообразие ротавируса А в Москве и на основании этих, а также литературных данных были определены наиболее представленные в центральной части России генотипы, с учетом первичной последовательности генов, кодирующих белки VP4 и VP7. Более 90 % образцов принадлежали к генотипам G9P[8], G4P[8], G2P[4], G1P[8]. Таким образом, для включения в состав ВПЧ были выбраны 6 генотипов белков внешнего слоя (G1, G2, G4, G9, P4, P8), а также белки VP2 и VP6, необходимые для сборки ВПЧ.
Для создания конструкции VP2/VP6, последовательность этих генов была оптимизирована для экспрессии в клетках насекомых, получена синтетическим путем, затем клонирована в соответствующие участки вектора pFastBacDual. Этот вектор предназначен для создания рекомбинантного бакуловируса, одновременно экспрессирующего два разных клонированных гена. Согласно литературным данным, добавление eGFP к VP2 не влияет на способность химерного белка участвовать в формировании ВПЧ, при этом контроль за наращиванием рекомбинантного белка в культуре клеток значительно упрощается возможностью визуального наблюдения за процессом в режиме реального времени с помощью флуоресцентного микроскопа.
Пример 1. Получение трехслойных вирусоподобных частиц, с использованием рекомбинантных бакуловирусов.
Нуклеотидные последовательности, кодирующие выбранные генотипы белков VP4 и VP7, были оптимизированы для наработки в клетках насекомых и получены синтетическим путем, затем клонированы в трансферный вектор pFastBacHTa. Особенностью данного вектора является наличие гена полигистидинового фрагмента на N-конце белка, для очистки продукта методом металлоаффинной хроматографии и детекции с применением специфических антигистидиновых МКА. Каждая последовательность была клонирована в отдельный трансферный вектор.
С помощью трансферных векторов гены белков ротавируса А были перенесены в бакуловирусный геном. После трансфекции клеток Sf-21 бакмидами, содержащими гены белков VP6 и eGFP-Delta92-VP2, следующий пассаж вируса проводили на клетках T. ni и наблюдали за появлением флуоресценции, сравнивая клетки, зараженные бакуловирусом дикого типа с клетками, зараженными eGFP-Delta92-VP2/VP6. Результаты представлены на фигуре 1 (Г-Е).
Кинетику накопления базовых VLP eGFP-Delta92-VP2/VP6 оценивали по флуоресценции на инвертированном микроскопе Olympus и методом непрямого ИФА с моноклональными антителами к полигистидину. В результате было показано, что максимальное накопление антигенов наблюдалось на вторые сутки после заражения. Однако в случае коинфицирования рекомбинантным бакуловирусом со вставкой VP2/VP6 с одним или двумя другими рекомбинантными бакуловирусами (VP4, VP7), происходит значительное снижение числа инфицированных клеток. Это может быть связано с интерференцией между вирусами. Снижение дозы бакуловирусов со вставкой VP4 и VP7 позволило решить эту проблему и добиться необходимого уровня экспрессии генов VP2/VP6 при коинфекции сопоставимого с таковым при моноинфекции (фиг 1 (Ж-И).
Таким образом, были подобраны оптимальные рекомбинантные бакуловирусы для каждой конструкции, а также длительность инкубации культуры клеток насекомых, инфицированных различными рекомбинантными бакуловирусами.
Полученные данные были использованы для проведения коинфекции, то есть одновременного заражения перевиваемой линии клеток насекомых T.ni различными сочетаниями рекомбинантных бакуловирусов.
Анализ структурных белков и чистоту VLP оценивали методом электрофореза в 12% полиакриламидном геле с додецилсульфатом натрия (ПААГ-ДСН) по методу Laemmli (1970) Электрофорез проводили в пластинах полиакриламидного геля размером 70 х 100 х 0,75 на приборе Mini-PROTEAN II (Bio-Rad, США) в восстанавливающих условиях при по-стоянном напряжении 200 V. Разделяющий гель содержал 12% акриламида, 0,5% N,N-метилен-бис-акриламида, 0,375 М трис-HCl pH 8,8 и 0,1% ДСН. Фокусирующий гель содержал 4% акриламида, 10% N,N-метилен- бисакриламида в 0,125 М трис-HCl буфере pH 6,8. Для полимеризации в оба геля вносили по 0,025% персульфата аммония и 0,075% TEMED. Электродный буфер содержал 0,025 М трис-HCl, 0,192 М глицина, pH 8,3 и 0,1% ДСН. Все испытуемые пробы содержали лизирующий буфер с восстановителем (0,125 М трис-HCl, pH 6,8, 5% ДСН, 0,5% β -меркаптоэтанола, 10,8% глицерина, 0,01% бромфенолового синего) и были прогреты в течение 5 минут при 100°. Заливку геля и подготовку аппарата для электрофореза к работе проводили согласно рекомендациям изготовителя. Белки в гелях окрашивали в течение 1 часа 0,1% раствором Кумасси ярко-голубого (CBB R-350) в водном растворе, содержащем 10% уксусной кислоты и 30% метанола. Избыток красителя отмывали 10% раствором уксусной кислотой такое же время с несколькими его сменами. В качестве белков-маркеров молекулярной массы использовали β-галактозидазу- 116 кД; фосфорилазу В - 94 кД, БСА- 66 кД, овальбумин- 45 кД, карбонат ангидразу-30 кД, ингибитор трипсина- 20,1 кД. На фигуре 2 показан пример исследования трехслойных VLP, содержащих VP2, VP6 и VP4.
Таким образом, было определено, что в результаты синтеза трехслойных частиц синтезируются три белка ротавируса.
Для оценки вирусного продукта VP6 проводили иммуноферментный анализ. Рекомбинантный белок VP6 был окрашен специфическими моноклональными антителами RG23B9С5Н11 (любезно предоставлены профессором Linda Saif, OSU). Поскольку белки VP2 и VP6 были клонированы в одной кассете, то экспрессия VP6 одновременно подтверждала экспрессию VP2.
Таким образом, был получен препарат, содержащий очищенные трехслойные VLP ротавирусов.
Пример 2. Очистка VLP ротавируса.
Культуральную жидкость, содержащую вирус или вирусоподобные частицы, подвергали низкоскоростному центрифугированию, освобождаясь от клеток и клеточного дебриса при 1000 об/мин в течение 5 минут и при 6000 об/мин в течение 20 минут соответственно (+4°С, ротор Sorval® SS34). После этого супернатант использовался для выделения и очистки ротавирусных-VLP методом ультрацентрифугирования.
Полученные осветлённые суспензии наслаивали на 6 мл 25% или 35% (w/v) сахарозы, приготовленной на буфере TNС (10 mM Tris-HCl, 140 mM NaCl, 10 mM CaCl2 рН 7.4). Центрифугировали в течение 2 часов при 28 000 об/мин (центрифуга Оptima XE-100, ротор SW 32Ti, Beckman Coulter, +4°С). Полученные осадки ресуспендировали в буфере TNС, и хранили при температуре +4°С.
Пример 3. Определение VLP методом электронной микроскопии
3 мкл препарата, содержащего VLP, с концентрацией 12-30 мг/мл наносили на медную сетку, покрытую углеродной подложкой (Ted Pella, США) и обработанную в атмосфере тлеющего разряда, инкубировали 30 секунд при комнатной температуре. Затем на сетку наносили каплю 2% раствора ацетата урана, выдерживали 30 секунд, излишки раствора удаляли с сетки фильтровальной бумагой. Окрашенные сетки хранили в пластиковых контейнерах до использования. Исследование образцов производили в просвечивающем электронном микроскопе JEOL 2100 (JEOL, Япония), оборудованном катодом из гексаборита лантана, при ускоряющем напряжении 200 кВ. Изображения получали с увеличением х25000 с помощью ПЗС-камеры Gatan X100 с размером матрицы 2000х2000 пикселей (Gatan, США). Вирусоподобные частицы, визуализированные методом электронной микроскопии представлены на фиг 4.
Пример 4. Определение специфической активности очищенных VLP методом ИФА.
Специфическую активность очищенных VLP оценивали методом ИФА. В качестве сорбированного антигена использовали МкАт к белку VP6 РВА. Очищенные VLP и РВА титровали с шагом ×3 в разведениях от 1/100 до 1/218700. Для выявления связавшихся с сорбированным антигеном VLP и РВА использовали меченные пероксидазой МкАт к белку VP6 РВА (фиг. 3). Результаты ИФА показали высокий титр очищенных VLP по сравнению с РВА, что свидетельствует об их высокой специфической активности.
Пример 5. Выбор способа введения и оптимизация адъюванта на модели мышей
Для исследования использовали самок мышей линии Balb/c 3-4 недельного возраста массой 16-18 г (питомник лабораторных животных Филиал «Столбовая» ФГБУН «НЦБМТ» ФМБА, Россия).
Проводили двукратную иммунизацию с интервалом 14 дней. При интраназальном введении применяли легких эфирный наркоз. В качестве адъюванта использовали адъювант на основе сквалена. Сформировали 7 опытных групп:
Группа № 1. Внутримышечное введение с адъювантом в объёме 0,2 мл/мышь.
Группа № 2. Внутримышечное введение без адъюванта в объёме 0,2 мл/мышь.
Группа № 3. Внутрибрюшинное введение с адъювантом в объёме 0,2 мл/мышь.
Группа № 4. Внутрибрюшинное введение без адъюванта в объёме 0,2 мл/мышь
Группа № 5. Интраназальное введение с адъювантом в объёме 0,05 мл/мышь.
Группа № 6. Интраназальное введение без адъюванта в объёме 0,05 мл/мышь.
Группа 7 - Контроль.
Через 7 дней после второй иммунизации от всех животных получали сыворотку крови и определяли уровень антител к белку VP6 и VLP РВА методом непрямого ИФА с использованием в качестве сорбированного антигена лизат клеток, содержащих рекомбинантный белок VP6 и суспензию VLP. Сыворотки тестировали с двойным шагом в разведениях от 1/200 до 1/25600. Использовали конъюгат анти-IgG мыши (Sigma A4416) в разведении 1/2000. Результаты представлены на фиг. 5.
Исследование способа введения и применение адъюванта показало, что все исследованные способы введения VLP в сочетании с адъювантом и без вызывают образование специфических IgG к белкe РВА VP6 и к VLP. Однако наибольший уровень антител наблюдается при внутримышечном введении с адъювантом. Учитывая дальнейшее применение вакцины против РВИ в клинической практике, в частности вакцинацию детей до 2-х лет, для дальнейшего исследования на релевантных видах животных были выбраны две формуляции прототипной вакцины против РВИ на основе VLP и VLP в комбинации с аттенутрованным штаммом РВА при внутримышечном и интраназальном способах введения.
Пример 6. Выбор дозы прототипных вакцин на модели новорожденных карликовых свиней
Для конечной формуляции прототипной вакцины на основе VLP - рекомбинантных белки: VP6-VP7 (генотип G1), VP6-VP7 (генотип G2), VP6-VP7 (генотип G4), VP6-VP7 (генотип G9), VP6-VP4 (генотип P4), VP6-VP4 (генотип P8), актуальных для эпидемических изолятов генотипов, наиболее распространенных в Российской Федерации, смешивали в равных количествах (по белку). Смесь VLP смешивали с адъювантом в соотношении 1:1.
В исследовании использовали клинически здоровых поросят возрастом от 5 до 6 дней от 5 супоросных свиноматок, показавших отрицательный результат на наличие специфических IgG к РВА.
Всего исследовали 10 групп животных, из которых шести группам вводили прототипные вакцины на основе VLP, двум - плацебо и ещё две группы были контрольными. Распределение по группам осуществляли в день первой иммунизации (Таблица 1).
30 мкг/доза с адъювантом
150 мкг/доза с адъювантом
750 мкг/доза с адъювантом
30 мкг/доза с адъювантом
150 мкг/доза с адъювантом
750 мкг/доза с адъювантом
Исследуемые кандидатные вакцины на основе VLP вводили животным внутримышечно и интраназально, как возможные пути применения в клинической практике. Введение осуществляли трехкратно, с интервалом 2 недели (на 0-й, 14-й и 28-й день эксперимента).
На 0-й, 14-й, 28-й, 42-й дни исследования у животных отбирали кровь в пробирки с активатором свертывания («Guangzhou Improve Medical Instruments Co»., Ltd, Китай, серия С31003, годен до 08.2022) в объеме 2,0 мл. Кровь центрифугировали (3000 об/мин, 15 минут, при комнатной температуре), полученную сыворотку использовали для выявления специфических антител методом ИФА и в РН. Результаты ИФА представлены на фиг. 6.
Исследование гуморального иммунитета.
Результаты определения специфических IgG к белку VP6 РВА показали, что уже после второй иммунизации животных прототипной вакциной на основе VLP внутримышечно уровень специфических антител резко возрастает и увеличивается после трехкратной иммунизации. При этом минимальная исследуемая доза VLP 30 мкг после трехкратной иммунизации вызывает значительное увеличение уровня специфических антител по сравнению с дозами 150 мкг и 750 мкг (фиг. 6).
Определение уровня вируснейтрализующих антител
Определение уровня вируснейтрализующих антител проводили на 28 и 42 день исследования. В результате установлено, что образование вируснейтрализующих антител наблюдается после второй иммунизации прототипной вакциной на основе VLP в группах №№ 1-3 во всех исследуемых дозах. При этом при иммунизации минимальной и максимальной исследуемыми дозами 30 мкг и 750 мкг соответственно, после третьей иммунизации титр нейтрализующих антител увеличивается вдвое (фиг 7).
Исследование Т-клеточного иммунитета
Клеточно-опосредованный иммунный ответ, индуцированного вакциной на основе VLP был исследован методом бласттрансформации лимфоцитов (РБТЛ).
Через 10 дней после первой иммунизации у всех животных (см. таблицу 1) отбирали периферическую кровь для исследовали и оценки уровня Т-клеточного иммунитета. На фигуре 8 представлены результаты реакции бласттрансформации лимфоцитов (РБТЛ) с нормированием на: нестимулированные моноциты, моноциты, стимулированные неспецифичным антигеном (ККГЛ), носитель, адъювант.
Результаты РБТЛ показали, что все исследуемые нами концентрации способны стимулировать пролиферацию моноцитов периферической крови поросят при внутримышечном введении VLP и интраназальном введении VLP. Сравнение значений ИСП в группах с внутримышечным способом введения (1-3) не показал статистически значимых различий в стимуляции пролиферации при минимальной (30 мкг/доза) и максимальной (750 мкг/доза) используемой концентрациях (ИСП 29,5±7 и ИСП 19,7±4,6, соответственно). При сравнении значений ИСП в группах с интраназальным способом введения (4-6) показано, что введение поросятам минимальной исследуемой дозы в группе 4 (30 мкг/доза) индуцирует наиболее интенсивную пролиферацию (ИСП 21,9±2,3) по сравнению с группами 5 (150 мкг/доза) и 6 (750 мкг/доза), p <0,05. Следует отметить, что в группах животных с внутримышечным и интраназальным введением суспензии VLP отмечаются наибольшие значения ИСП 29,5±7 и ИСП 21,9±2,3, соответственно, в минимальной исследуемой концентрации (группа 1 и 4) и значения ИСП не различаются статистически значимо, p >0,05.
Полученные результаты свидетельствуют о специфическом клеточном ответе у всех исследуемых иммунизированных животных после первой иммунизации, как при интраназальном, так и внутримышечном введении суспензии VLP.
Таким образом, представленные примеры показывают достижение заявленного технического результата и промышленную применимость заявленного изобретения.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing originalFreeTextLanguageCode="ru"
dtdVersion="V1_3" fileName="ВПЧ Ротавирус А.xml" softwareName="WIPO
Sequence" softwareVersion="2.2.0" productionDate="2022-12-05">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText></ApplicationNumberText>
<FilingDate></FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>1</ApplicantFileReference>
<ApplicantName languageCode="ru">федеральное государственное
бюджетное учреждение «Национальный исследовательский центр
эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи»
Министерства здравоохранения Российской Федерации</ApplicantName>
<ApplicantNameLatin>federalnoe gosudarstvennoe biudzhetnoe
uchrezhdenie Natsionalnyi issledovatelskii tsentr epidemiologii i
mikrobiologii imeni pochetnogo akademika N F Gamalei Ministerstva
zdravookhraneniia Rossiiskoi Federatsii</ApplicantNameLatin>
<InventionTitle languageCode="ru">ИММУНОБИОЛОГИЧЕСКОЕ СРЕДСТВО НА
ОСНОВЕ ВИРУСОПОДОБНЫХ ЧАСТИЦ ДЛЯ ИНДУКЦИИ СПЕЦИФИЧЕСКОГО ИММУНИТЕТА
ПРОТИВ ИНФЕКЦИИ, ВЫЗЫВАЕМОЙ РОТАВИРУСОМ А ЧЕЛОВЕКА</InventionTitle>
<SequenceTotalQuantity>8</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>3185</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..3185</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctcgagatggtgtccaagggagaggagctgttcacgggagtggtgccaa
ttctggtggagctggacggagacgtgaacggccacaagttcagcgtgagcggagagggagaaggagacgc
cacatacggcaagctgaccctgaagttcatttgcaccaccggaaaattgcccgtgccctggccaacactg
gtgacaacactgacctacggcgtgcagtgcttcagccgttaccccgaccacatgaagcagcacgacttct
tcaagagcgccatgcccgagggatacgtgcaggagcgcaccatcttcttcaaggacgacggcaactacaa
gacccgcgccgaagtgaagttcgagggagacaccctggtgaaccgcatcgagctgaagggcatcgacttc
aaggaggacggcaacatcctgggccacaagctggagtacaactacaacagccacaacgtgtacatcatgg
ccgacaagcagaagaacggcatcaaggtcaacttcaagatccgccacaacatcgaggacggaagcgtgca
atggccgaccactaccagcagaacaccccaatcggcgacggcccagtgctgctgccagataatcactacc
tgagcacccaaagcgccctgagcaaggatcccaacgagaagcgcgaccacatggtgctgctggagttcgt
gacagccgccggaattaccctgggcatggacgagctgtacaagagcggactgcgtagtcgagcccaggcc
agcaactccgctgtggatggtacagctggtccaggaagcacaggaagccgtagccgaggaagcaagacca
tcccaaccttcgagcccaaggagagcatcctgaagaagctggaggacatcaagcccgagcaggccaagaa
gcagaccaagctgttccgcatcttcgaaccacgccagctgccaatctatcgcgctaacggagagaaggag
ctgcgcaaccgttggtattggaagctgaagaaggacaccctgccggatggagattacgacgtgcgagagt
acttcctgaacctgtacgaccaggtgctgaccgagatgccagactacctgctgctgaaggacatggccgt
ggagaacaagaactcgcgcgatgccggaaaggtggtggatagcgaaaccgccagcatttgcgacgccatc
ttccaagatgaggaaacagaaggagccgtgcgccgtttcatcgccgaaatgcgacagcgcgtgcaggccg
atcgaaacgtggtgaactaccccagcatcctgcaccccatcgactacgccttcaacgagtacttcctgca
gcaccagctggtggagcccctgaacaacgacatcatcttcaactacatcccagagcgcatccgcaacgac
gtgaactacatcctgaacatggaccgcaacctgccaagtaccgcccgctacattcgcccaaacctgctgc
aggatcgcctgaacctgcacgacaacttcgagagcctgtgggacaccatcaccaccagcaactacatcct
ggcccgtagcgtggtgccagatctgaaggagctggtgtccaccgaggcccagatccagaagatgagccag
gacctgcagctggaagccctgaccattcagagcgagacccagttcctgaccggcatcaacagccaggccg
ccaacgattgcttcaagaccctgatcgccgccatgctgagtcaacgtaccatgagcctggacttcgtgac
caccaactacatgagcctgatctccggcatgtggttgctgaccgtggtgcccaacgacatgttcatccgc
gagagcctggtcgcttgccaactggccatcgtgaacaccatcatctaccccgccttcggcatgcagcgaa
tgcactaccgcaacggcgatccacagcgtccattccaaatcgccgagcagcagatccagaacttccaagt
ggccaattggctgcacttcgtgaacaacaaccagttccgccaggtggtgatcgacggagtgctgaaccag
gtgctgaacgacaacatccgcaacggccacgtgatcaaccagctgatggaggccctgatgcagctgagtc
gccagcagttcccaaccatgccagtggactacaagcgcagcatccagcgaggcatcctgttgctgagcaa
tcgcctgggacagctggtggatctgacccgactgctggcctacaactacgagaccctgatggcctgcgtc
accatgaacatgcagcacgtgcagaccctgaccaccgaaaagctgcagctgaccagcgtgaccagcttgt
gcatgctgatcggcaacgccaccgtgatcccaagcccccagaccctgttccactactacaacgtgaacgt
gaacttccacagcaactacaacgagcgcatcaacgacgccgtggctatcatcaccggagccaaccgcctg
aacctgtaccagaagaagatgaaggccatcgtggaggacttcctgaagcgcctgcacatcttcgacgtgg
cccgcgtgcccgacgatcagatgtaccgtctgcgagatcgtctgcgactgttgccagtggaagtgcgccg
cctggacatcttcaacctgatcctgatgaacatggaccagatcgagcgcgccagcgataagattgcccaa
ggagtgatcatcgcctaccgcgacatgcagctggagcgagacgagatgtacggctacgtgaacatcgccc
gcaatctggacggattccaacagatcaacctggaggagctgatgcgcaccggagattacgcccagatcac
caacatgctgctgaacaatcagccagtggccctggtgggagctttgcccttcgtgaccgatagcagcgtg
atcagcctgatcgccaacgtggacgctaccgtcttcgcccagatcgtgaagctgcgaaaggtggacaccc
tgaagcccatcctgtacaagatcaacagcgacagcaacgacttctacctggtggccaactacgattgggt
gcccaccagcaccaccaaggtgtacaagcaggtgccccagcagttcgacttccgcaacagcatgcacatg
ctgaccagcaacctgaccttcaccgtgtacagcgacctgctggctttcgtgtccgccgatacagtggagc
caatcaacgccgtggccttcgacaacatgcgcatcatgaacgagctgtaggctagc</INSDSeq_sequ
ence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>1234</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..1234</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggatccatggaagtcctgtactccttgagcaagactctgaaagatgccc
gtgacaagatcgtggaaggcactctctactccaacgtgtctgacctgatccagcagttcaatcagatgat
cgtgaccatgaatggaaacgactttcagactggtggcattggcaaccttcctatcaggaactggaccttt
gactttggtctccttggcactacactgttgaatctggatgccaattacgttgagaccgccaggactacca
ttgagtacttcatcgacttcatcgacaacgtgtgcatggatgagatggcccgtgagtctcagcgcaatgg
tgtcgctccacagtctgaagctctgcgcaaacttgctggcatcaagttcaagcgcatcaacttcaacaac
agctctgagtatatcgagaactggaacttacagaacagacgtcaacgcactggcttcgtgtttcacaaac
ccaacatctttccatactctgcctccttcaccttgaacagatctcaacctatgcacgacaacctcatggg
aaccatgtggctgaatgctggctctgagattcaagttgctggctttgctggctctgagattcaagttgct
ggctttgactacagctgtgcactgaatgctcctgccaatattcaacagtttgaacacatcgtccaactgc
gtagagccttgaccactgccaccatcactctgttgcctgatgctgagcgcttcagcttccctagagtgat
caactctgctgatggtgccactacatggttcttcaatcccatcattctgagacccaacaatgtggaagtt
gagttcttgctgaatggtcagatcattaacacctaccaagctaggtttggcaccatcattgctcgcaact
ttgacaccattcgtctgtccttccaactgatgagacctcccaacatgactccagctgtgaatgctctctt
tcctcaagctcaaccctttcaacaccatgccaccgttggtctgactcttcgcatcgagtctgctgtgtgt
gaatcagtgcttgccgatgccaatgagaccttgcttgctaatgtgactgctgttcgtcaagagtatgcca
ttccagttggtcctgtgtttccacctggcatgaactggactgaactgatcaccaactactctccttcccg
tgaagacaacttgcaaagagtcttcaccgttgccagcatccgtagcatgttgatcaagtaagctt</INS
DSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>993</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..993</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gaattcatgtacggcatcgagtacaccaccatcctgatcttcctgatca
gcatcatcctgctgaactacatcctgaagagcgtgacccagatgatggactacatcatctaccgcagcct
gctgatcagtgtggccctgtttgccctgacacgagcccagaactacggcatcaacctgcccatcaccgga
agcatggacgccgcttacgccaatagcacccaggagggcatattcctgaccagcaccctgtgcctgtact
acccaaccgaggctagcacccagatcaacgacggcgagtggaaggacagcctgagccagatgttcctgac
caagggttggcccaccggaagcgtgtacttcaaggagtactccagcatcgtggagttcagcgtggacccc
cagctgtattgcgactacaacctggtgctgatgaagtacgaccagaacctggagctggacatgagcgagc
tggccgatctgatcctgaacgagtggctgtgcaaccccatggacatcaccctgtactactaccagcagag
cggcgagagcaacaagtggatcagcatgggtagctcctgcaccgtgaaggtctgcccactgaacacccag
acactgggaatcggttgccagaccaccaacgtggacagcttcgagatggtggccgagaacgagaagctgg
ccatcgtggacgtcgtggacggcatcaaccacaagatcaacctgaccaccaccacttgcaccatccgcaa
ttgcaagaagctgggaccacgcgagaacgtggctatcatccaggtcggaggcagcaacgtgctggatatc
accgccgatcccaccaccaatccacagaccgagcgcatgatgcgcgtgaattggaagaagtggtggcagg
tgttctacaccatcgtggactacatcaaccagatcgtgcaggtcatgagcaagcgcagtcgcagtctgaa
tagcgccgccttctactatcgcgtgtgagagctc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>993</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..993</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gaattcatgtacggcatcgagtacaccaccatcctgaccatcctgatct
tcatcatcctgctgaactacatcctgaagaccatcaccaacaccatggactacatcatcttccgcttcct
gctgctgatcgccctgatgagtccattcgtgcgcacccagaactacggcatgtacctgcccatcaccgga
agcctggacgccgtgtataccaacagcaccagcggagagagctttctgaccagcaccctgtgcctgtact
acccaaccgaggccaagaacgagatcagcgacaacgagtgggagaacaccctgagccagctgttcctgac
caagggttggccaaccggcagcgtgtacttcaaggactacaacgacatcaccaccttcagcatgaacccc
cagctgtactgcgactacaacgtggtcctgatgcgctacgacaacaccagcgagctggacgctagcgagc
tggccgatctgattctgaacgagtggctgtgcaaccccatggacatcagcctgtactactaccagcagaa
cagcgagagcaacaagtggatcagcatgggtaccgactgcaccgtgaaggtctgcccactgaacacccag
accctgggaatcggttgcaagaccaccgacgtggacaccttcgagatcgtggctagcagcgagaagctgg
tgatcaccgacgtggtgaacggcgtgaaccacaagatcaacatcagcatcagcacttgcaccatccgcaa
ttgcaacaagctgggaccacgcgagaacgtggctatcatccaagtgggcggcccaaacgctctggatatc
accgccgatccaaccaccgtgccacaagtgcagcgcatcatgcgcgtgaattggaagaagtggtggcagg
tgttctacaccgtggtggactacatcaaccagatcatccaggtcatgagcaagcgcagtcgcagtctgga
tacagccgccttctactaccgcatctgagagctc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>996</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..996</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gaattcatgtacggcatcgagtacaccgccgtgctgttctacctgatca
gcttcgtgttcgtgtcctacatcctgaagaccatcatcaagatcatggactacatcatctaccgcatcac
cttcgtgatcgtcgtgctgagcgtgctgagtaacgcccagaactacggcatcaacctgcccatcaccgga
agcatggataccgcctacgccaacagcacccaggacaacaacaacttcctgagcagcaccctgtgcctgt
actacccaagcgaggctccaacccagatcagcgacaccgagtggaaagacaccctgagccagctgttcct
gactaagggctggccaaccggaagcgtgtacttcaacgagtactccaacgtgctggagttcagcatcgag
cccaagctgtattgcgactacaacgtcgtgctgatcaagttcaccagcggcgaggagctggacatcagcg
aactggccgacctgatcctgaacgagtggctgtgcaaccccatggacatcaccctgtactactaccagca
gaccggcgaggccaacaagtggatcagcatgggcagcagttgcaccgtgaaggtctgccccctgaacaca
cagaccctgggaatcggttgccagaccaccaataccgccaccttcgagacagtggccgacagcgagaagc
tggccatcatcgacgtcgtggacagcgtgaaccacaagctggacgtgaccagcaccacttgcaccatccg
caactgcaacaagctgggaccacgcgagaacgtggctatcatccaggtcggaggcagcaacgtgctggat
atcaccgccgatcccaccaccagtccacagaccgagcgcatgatgcgcgtgaattggaagaagtggtggc
aggtgttctacaccgtggtggactacatcaaccagatcgtgcaggtgatgagcaagcgcagccgtagcct
ggatagcagcagcttctactaccgcgtgtgagagctc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>993</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..993</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gaattcatgtacggcatcgagtacaccaccatcctgaccttcctgatca
gcatcgtgctgctgaactacatcctgaagagcctgaccagcgccatggacttcatcatctaccgcttcct
gctgctgatcgtgatcgccagccccttcgtgaagacccagaactacggcatcaacctgcccatcaccggc
agcatggataccgcttacgccaacagcagccagcaggaaaccttcctgaccagcaccctgtgcctgtact
acccaaccgaggctagcacccagatcggcgataccgagtggaaggacaccctgagccagctgttcctgac
caagggttggccaaccggcagcgtgtacttcaaggagtacaccgacatcgccagcttcagcatcgacccc
cagctgtattgcgactacaacgccgtgctgatgaagtacgacagcaccctggagctggacatgagcgagc
tggccgatctgatcctgaacgagtggctgtgcaaccccatggacatcaccctgtactactaccagcagac
cgacgaggccaacaagtggatcagcatgggtcagagttgcaccatcaaggtctgccccctgaacacccag
accctgggaatcggttgcatcaccaccaacaccgccaccttcgaggaagtggctacctccgagaagctgg
tgatcaccgacgtggtggacggagtgaaccacaagctggacgtgaccaccaacacttgcaccatccgcaa
ttgcaagaagctgggaccacgcgagaacgtggctatcgtgcaagtgggaggcagcaacgtgctggatatc
accgccgatccaaccaccacaccacagaccgagcgcatgatgcgcgtgaattggaagaagtggtggcagg
tgttctacaccgtggtggactacatcaaccagatcgtgcaggtcatgagcaagcgcagtcgcagtctgaa
tagcgccgccttctactatcgcgtgtgagagctc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>2340</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..2340</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gaattcatggctagcctgatctaccgccagctgctgaccaacagctaca
gcgtggacctgcacgacgagatcgagcagatcggcagcgagaagacccagaacgtgaccgtgaaccccgg
accattcgctcagacacgctacgccccagtgaattggggacacggcgagatcaacgatagcaccaccgtg
gagccagtgttggacggaccataccagccaaccaccttcaagccccccaacgactactggctgctgatca
gcagcaacacagacggcgtggtgtacgagagcaccaacaacagcgacttttggaccgccgtgatcgccgt
ggaaccacacgtgtcccagacaaaccgccgttacgtgctgttcggcgagaacaagcagttcaacatcgag
aacagcagcgacaagtggaagttctttgagatgttcaagggcagcagccagagcgacttcagcaaccgcc
gcaccctgacaagcgataaccgcctggtgggcatgctgaagtacggaggacgcgtctggaccttccacgg
tgaaacaccacgcgctaccaccgatagcagcaataccgccgacctgaacaacatcagcatcatcatccac
agcgagttctacatcatcccccgcagccaggagagcaagtgcaacgagtacatcaacaacggcctgccac
caatccagaatacccgcaatgtggtgccactgagcctgagcagtcgcagcatccagtaccgtcgcgccca
agtgaacgaggacatcaccatcagcaagaccagcctgtggaaggagatgcagtacaaccgcgacatcatc
atccgcttcaagttcggcaacagcgtgatcaagctgggcggactgggctacaagtggagcgagatcagct
acaaggccgccaactaccagtacagctacagccgcgacggagaacaggtgacagcccacaccacctgcag
cgtgaacggcgtgaacaacttcagctacaacggcggcagcctgccaaccgatttctccatcagccgctac
gaggtcatcaaggagaacagctacgtgtacatcgactattgggacgacagcaaggccttccgcaacatgg
tctacgtgcgcagcctggccgctaacctgaacagcgtgaagtgcttgggcggaagctacgatttccgact
gccagtgggcgagtggccaattatgaacggaggagccgtgtccctgcattttgccggagtgaccctgagc
acccagttcaccgacttcgtgtccctgaacagcctgcgctttcgcttcagcctgaccgtggacgagccaa
gcttcagcatcatccgcacccgcaccatgaacctgtacggactgccagccgctaaccccaacaacggcaa
cgagtactacgaggtgtccggccgcttcagcctgatcagcctggtgccaaccaacgacgactaccagacc
cccatcatgaacagcgtgaccgtgcgacaggatctggagcgacagctgaacgacctgcgagaggagttca
acagcctgagccaggagatcgccatgagccagctgatcgatctggccttgctgcccctggacatgttctc
gatgttctccggcatcaagagcaccatcgacctgaccaagagcatggccaccagcgtgatgaagaagttc
cgcaagagcaagctggccaccagcatcagcgagatgaccaacagtctgagcgacgccgctagcagcgcta
gccgaagcgctagcattcgcagcaacctgagcaccatcagcaactggtcggatgccagcaagagcgtgct
gaacgtgaccgacagcgtgaacgacatcagcacccagaccagcaccatcagcaagaagctgcgcctgaag
gagatgatcacccagaccgagggcatcagcttcgacgatatttccgccgccgtgctgaagaccaagatcg
acatgagcacccagatcggcaagaacaccctgccagacatcgtgaccgaggctagcgagaagttcatccc
caagcgcagctaccgcgtgttgaaggacgacgaggtgatggaggtcaacaccgagggcaagttcttcgcc
tacaaggtggacaccctgaacgagatccccttcgacatcaacaagttcgccgagctggtgaccgatagcc
cagtgatcagcgccatcatcgacttcaagaccctgaagaacctgaacgacaactacggaatcacccgcat
cgaggccctgaacctgatcaagagcaaccccaacgtgctgcgcaacttcatcaaccagaacaaccccatc
atccgcaaccgcatcgagcagctgatcctgcagtgcaagctgtgagagctc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>2340</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..2340</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q16">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gaattcatggctagcctgatctaccgccagctgctgaccaacagctaca
gcgtggacctgcacgacgagatcgagcagatcggcagcgagaagacccagaacgtgaccgtgaaccccgg
accattcgctcagacacgctacgccccagtgaattggagccacggagagatcaacgacagcaccaccgtg
gagccaatcctggacggaccataccagccaaccaccttcaccccccccaccgattattggatcctgatca
ccagcaacaccaacggcgtggtgtacgagagcaccaacaacagcgacttttggaccgccgtgatcgccgt
ggaaccacacgtgaacccagtggaccgccagtacaacgtgttcggcgagaacaagcagttcaacgtgcgc
aacgacagcgacaagtggaagttcttggagatgttccgcggcagcagccagaacgatttctacaaccgcc
gcaccctgaccagcgatacacgcctggtgggcatcctgaagtacggaggacgcgtctggaccttccacgg
tgaaacaccacgcgctaccaccgatagcagcaataccaccaacctggacggcatcagcatcaccatccac
agcgagttctacatcatcccccgcagccaggagagcaagtgcaacgagtacatcaacaacggcctgccac
caatccagaatacccgcaatgtggtgccactgagcctgagcagtcgcagcatccagtacaagcgcgccca
ggtcaacgaggacatcaccatcagcaagaccagcctgtggaaggagatgcagtacaaccgcgacatcatc
atccgcttcaagttcggcaacagcatcgtgaagctgggaggactgggctacaagtggagcgagatcagct
acaaggccgccaactaccagtacaactacctgcgcgacggcgagcaagtgacagcccacaccacctgcag
cgtgaacggcgtgaacaacttcagctacaacggcggcagtctgccaaccgatttctccgtgtcccgctac
gaggtcatcaaggagaacagctacgtctacgtggactattgggacgacagcaaggccttccgcaacatgg
tctacgtgcgcagcctggccgctaacctgaacagcgtgaagtgcacgggaggcagctacaacttcagcat
ccccgtgggagcttggccagtgatgaacggaggagccgtgtccctgcattttgccggagtgaccctgagc
acccagttcaccgacttcgtgtccctgaacagcctgcgcttccgctttagcctgaccgtggatgagccca
gcttcagcatcctgcgcacccgcacagtgaacctgtacggactgccagccgctaaccccaacaacggcaa
cgagtactacgagatcagcggccgcttcagcctgatcagcctggtgccaaccaacgacgactaccagacc
cccatcatgaacagcgtgaccgtgcgacaggatctggaacgacagctgaccgacctgcgagaggagttca
acagcctgagccaggagatcgccatgagccagctgatcgatctggccttgctgcccctggacatgttctc
gatgttctccggcatcaagagcaccatcgacctgaccaagagcatggccaccagcgtgatgaagaagttc
cgcaagagcaagctggccacaagcgtgtccgagatgaccaacagtctgagcgacgccgctagcagcgcta
gccgtagcgtgtccgtgcgcagcaacatcagcgccatcagcaactggaccaacgtgtccaacgacgtctc
caacgtgaccaacagcgtgaacgacatcagcacccagaccagcaccatcagcaagaacctgcgcctgaag
gagatgatcacccagaccgagggcatgagcttcgacgatatttccgccgccgtgctgaagaccaagatcg
acatgagcacccagatcggcaagaacaccctgccagacatcgtgaccgaggctagcgagaagttcatccc
caagcgcagctaccgcatcctgaaggacgacgaggtgatggagatcaacaccgagggcaagttcttcgcc
tacaagatcgacaccctgaacgaggtgcccttcgacgtgaacaagttcaccgagctggtgaccaatagcc
ccgtgatcagcgccatcatcgacttcaagaccctgaagaacctgaacgacaactacggaattacccgcac
cgaggccctgaacctgatcaagagcaaccccaacgtgctgcgcaacttcatcaaccagaacaaccccatc
atccgcaaccgcatcgagcagctgatcctgcagtgcaagctgtgagagctc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Вакцина против ротавирусной инфекции человека на основе вирусоподобных частиц ротавируса | 2024 |
|
RU2829862C1 |
| Вирусоподобные химерные частицы для индукции специфического иммунитета против вируса тяжелого острого респираторного синдрома SARS-CoV-2, содержащие белки коронавируса и ротавируса | 2022 |
|
RU2779810C1 |
| ВАКЦИНА ДЛЯ ПРОФИЛАКТИКИ И ЛЕЧЕНИЯ РОТАВИРУСНОЙ ИНФЕКЦИИ, СОДЕРЖАЩАЯ ГИБРИДНЫЙ БЕЛОК В КАЧЕСТВЕ АКТИВНОГО АГЕНТА (ВАРИАНТЫ) | 2013 |
|
RU2539913C2 |
| Вакцина на основе вирусоподобных частиц (VLP) для профилактики COVID-19 для парентерального применения | 2024 |
|
RU2834060C1 |
| Вакцина на основе вирусоподобных частиц (VLP) для профилактики COVID-19 для интраназального применения | 2024 |
|
RU2828323C1 |
| Рекомбинантные вирусоподобные частицы для индукции специфического иммунитета против вируса тяжелого острого респираторного синдрома SARS-CoV-2 | 2021 |
|
RU2769224C1 |
| РОТАВИРУСНЫЕ ЧАСТИЦЫ С ХИМЕРНЫМИ ПОВЕРХНОСТНЫМИ БЕЛКАМИ | 2014 |
|
RU2698049C2 |
| Способ получения активного начала вакцины против ротавирусной инфекции белка FliCVP6VP8 | 2017 |
|
RU2649132C1 |
| Способ определения генотипа ротавирусов первой и второй геногрупп и реассортантов между ними методом мультиплексной ПЦР | 2024 |
|
RU2833073C1 |
| РЕАССОРТАНТНЫЕ BTV И AHSV ВАКЦИНЫ | 2013 |
|
RU2656187C2 |

Изобретение относится к области биотехнологии и генной инженерии. Описано иммунобиологическое средство на основе вирусоподобных частиц ротавируса А. Средство содержит адъювант и капсидные гены ротавируса (VP2 и VP6), а также поверхностные белки VP4 и VP7 разных генотипов ротавируса, а именно G1, G2, G4, G9, P4, P8, синтезированные в бакуловирусной системе экспрессии и собранные в трехслойные вирусоподобные частицы (VLP). Изобретение обеспечивает создание средства, обладающего высокой степенью безопасности (как белковый препарат) и специфической иммуногенностью и эффективностью для профилактики ротавирусной инфекции, вызываемой определенными генотипами ротавируса А. 3 з.п. ф-лы, 8 ил., 1 табл., 6 пр.
1. Иммунобиологическое средство для индукции специфического иммунитета против инфекции, вызываемой ротавирусом А человека, содержащее адъювант и несколько рекомбинантных вирусоподобных трехслойных частиц, состоящих из двух белков капсидных генов ротавируса VP2 и VP6, имеющих нуклеотидные последовательности SEQ ID NO: 1, SEQ ID NO: 2, и одного или нескольких из поверхностных белков VP4 и VP7 различных генотипов ротавируса G1, G2, G4, G9, P4, P8, имеющих нуклеотидные последовательности SEQ ID NO: 3, SEQ ID NO: 4, SEQ ID NO: 5, SEQ ID NO: 6, SEQ ID NO: 7, SEQ ID NO: 8, которые получены в бакуловирусной системе экспрессии насекомых, при этом вирусоподобная частица имитирует вирион ротавируса А.
2. Иммунобиологическое средство по п.1, где адъювант представляет собой масляный адъювант.
3. Иммунобиологическое средство по любому из пп.1, 2, где адъювант представляет собой водно-масляную эмульсию.
4. Иммунобиологическое средство по любому из пп.1-3, где адъювант представляет собой водно-масляную эмульсию на основе сквалена.
| WO 2012049366 A1, 19.04.2012 | |||
| Вирусоподобные химерные частицы для индукции специфического иммунитета против вируса тяжелого острого респираторного синдрома SARS-CoV-2, содержащие белки коронавируса и ротавируса | 2022 |
|
RU2779810C1 |
| Пишущая машина с электрической передачей | 1932 |
|
SU34564A1 |
| ВАКЦИНА ДЛЯ ПРОФИЛАКТИКИ И ЛЕЧЕНИЯ РОТАВИРУСНОЙ ИНФЕКЦИИ, СОДЕРЖАЩАЯ ГИБРИДНЫЙ БЕЛОК В КАЧЕСТВЕ АКТИВНОГО АГЕНТА (ВАРИАНТЫ) | 2013 |
|
RU2539913C2 |
Авторы
Даты
2023-04-28—Публикация
2022-12-28—Подача