Изобретение относится к области молекулярной биологии, вирусологии и молекулярно-генетической диагностики, касается способа генотип-специфичной амплификации кДНК ротавирусов методом мультиплексной полимеразной цепной реакции и может быть использовано для дифференциации ротавирусов первой и второй геногрупп и выявления межгеногрупповых реассортантов.
Ротавирус А (вид Rotavirus А, семейство Sedoreoviridae) является возбудителем острого гастроэнтерита у детей первых лет жизни во всем мире. В России в последние 10 лет показатель заболеваемости ротавирусной инфекцией возрос более чем в 8 раз (с 9,3 до 80,9 на 100 тыс. населения) [Черепанова Е.А., Симонова Е.Г., Раичич P.P., Линок А.В., Филатов Н.Н. Оценка эпидемиологического риска в системе надзора за актуальными для Российской Федерации острыми кишечными инфекциями // Здоровье населения и среда обитания. - 2018. - №3 - С. 23-28]. Большое разнообразие генетических вариантов, способность к накоплению точечных мутаций и реассортации сегментов генома являются характерными чертами ротавируса A [Estes М.K., Greenberg Н.В. Rotaviruses. In: Knipe D.M., Howley P.M., eds. Fields Virology. Philadelphia: Lippincott Williams & Wilkins; 2013: 1347-401] и обуславливают необходимость постоянного мониторинга циркулирующих штаммов.
Ротавирусные частицы образованы тремя концентрическими слоями белков. Два белка наружного капсида, VP4 и VP7, вовлекаются в начальное взаимодействие вируса с клеткой-хозяином. Белок VP4 образует шипы, которые простираются от поверхности вирусной частицы, и участвует в прикреплении и проникновении в клетку, в то время как белок VP7 представляет собой гликопротеин, связывающий кальций, который образует гладкую поверхность вириона и предположительно участвует в процессе, происходящем после прикрепления [Mendez Е., Lopez S., Cuadras М.А., Romero P., Arias C.F. Entry of rotaviruses is a multistep process // Virology. - 1999. Vol. 263. №2. - P. 450-459].
Геном ротавирусов сегментирован, и это свойство обуславливает их способность к реассортации сегментов генома при одновременном инфицировании клетки двумя или более штаммами, в результате которой вирусное потомство получает смешанный набор родительских генов. Для описания разнообразия ротавирусов были разработаны две классификации - бинарная и полногеномная. Наиболее распространенная, бинарная классификация, базируется на двух генах, кодирующих белки наружного капсида ротавирусов VP7 и VP4 (G- и Р-генотипы). Для идентификации G- и Р-генотипа ротавируса созданы лабораторные методики на основе мультиплексной ПЦР в разных вариантах. Полногеномная классификация учитывает особенности всех 11 сегментов генома ротавирусов. В этом случае разными методами секвенируют полные гены или их фрагменты и затем, на основе нуклеотидной последовательности, идентифицируют полные генотипы с помощью онлайн-ресурсов. Использование этой классификации позволило четко разделить ротавирусы А на две геногруппы. В ранних исследованиях было показано, что внутренние гены (VP1, VP2, VP3, VP6, NSP1, NSP2, NSP3, NSP4, NSP5/6) ротавирусов человека генотипов G1P[8], G3P[8], G4P[8] и G9P[8] почти без исключений относятся к генотипу 1 и принадлежат первой или Wa-подобной геногруппе [Matthijnssens J., Ciarlet М., Heiman Е., Arijs I., Delbeke Т., McDonald S.M., et al. Full genome-based classification of rotaviruses reveals a common origin between human Wa-like and porcine rotavirus strains and human DS-1-like and bovine rotavirus strains // J. Virol. - 2008. Vol. 82. №7. - P. 3204-3219]. Их полный генотип обозначают следующим образом: Gl/3/4/9-P[8]-I1-R1-C1-M1-A1-N1-T1-E1-H1. Внутренние гены штаммов генотипа G2P[4], инфицирующих людей, как правило, относятся к генотипу 2 и принадлежат второй или DS-1-подобной геногруппе [Jere K.С., Mlera L., O’Neill H.G., Pot- gieter A.C., Page N.A., Seheri M.L., et al. Whole genome analyses of African G2, G8, G9, and G12 rotavirus strains using sequence-independent amplification and 454(R) pyrosequencing // J. Med. Virol. - 2011. Vol. 83. №ll. - P. 2018-2042.; Heiman E.M., McDonald S.M., Barro M., Taraporewala Z.F., Bar-Ma- gen Т., Patton J.T. Group A human rotavirus genomics: evidence that gene constellations are influenced by viral protein interactions / J. Virol. - 2008. Vol. 82. №22. - P. 11106-11116.; McDonald S.M., Matthijnssens J., McAllen J.K., Hine E., Overton L., Wang S., et al. Evolutionary dynamics of human rotaviruses: balancing reassortment with preferred genome constellations // PLoS Pathog. - 2009. Vol. 5.№10: el000634]. Их полный генотип обозначают как G2P[4]-I2-R2-C2-M2-A2-N2-Т2-Е2-Н2. Однако в дальнейшем во многих странах были обнаружены межгеногрупповые реассортанты, имеющие смешанный набор генов двух геногрупп. Например, начиная с 2011 г. в Японии, Таиланде и Вьетнаме были выявлены штаммы генотипа Gl-P[8]-I2-R2-C2-M2-A2-N2-T2-E2-H2, имеющие гены белков наружного капсида 1-й геногруппы и внутренние гены 2-й геногруппы ротавирусов [Kuzuya М., Fujii R., Hamano М., Kida K., Mizoguchi Y., Kanadani Т., et al. Prevalence and molecular characterization of G1P[8] human rotaviruses possessing DS-1-like VP6, NSP4, and NSP5/6 in Japan. J. Med. Virol. 2014; 86(6): 1056-64; Fujii Y., Nakagomi Т., Nishimura N., Noguchi A., Miura S., Ito H., et al. Spread and predominance in Japan of novel G1P[8] doublereassortant rotavirus strains possessing a DS-1-like genotype constellation typical of G2P[4] strains // Infect. Genet. Evol. - 2014. Vol.28. - P. 426 - 433.; Yamamoto S.P., Kaida A., Kubo H., Iritani N. Gastroenteritis outbreaks caused by a DS-1-like G1P[8] rotavirus strain, Japan, 2012-2013 // Emerg. Infect. Dis. - 2014. Vol. 20. - №6 - P. 1030-1033.; Komoto S., Tacharoenmuang R., Guntapong R., Ide Т., Haga K., Katayama K., et al. Emergence and characterization of unusual DS-1-Like G1P[8] rotavirus strains in children with diarrhea in Thailand // PLoS One. - 2015. Vol. 10. №11.: e0141739.; Phan M.V.T., Anh P.H., Cuong N.V., Munnink B.B.O., van der Hoek L., My P.T., et al. Unbiased whole-genome deep sequencing of human and porcine stool samples reveals circulation of multiple groups of rotaviruses and a putative zoonotic infection // Virus Evol. - 2016. 2(2).; Nakagomi Т., Nguyen M.Q., Gauchan P., Agbemabiese C.A., Kaneko M., Do L.P., et al. Evolution of DS-1-like G1P[8] double gene reassortant rotavirus A strains causing gastroenteritis in children in Vietnam in 2012/2013 // Arch. Virol. - 2017. Vol. 162. №3. - P. 739-48]. Также в качестве примера можно привести ротавирусы с генными констелляциями G3P[8]-I2-R2-C2-M2-A2-N2-T2-E2-H2 и G8-P[8]-I2-R2-C2-M2-A2-N2-T2-E2-H2, выявленные в Австралии, Испании, Японии, Таиланде и Вьетнаме [Tacharoenmuang R., Komoto S., Guntapong R. et al. Full Genome Characterization of Novel DS-1-Like G8P[8] Rotavirus Strains that Have Emerged in Thailand: Reassortment of Bovine and Human Rotavirus Gene Segments in Emerging DS-1-Like Intergenogroup Reassortant Strains // PLoS One. - 2016. - Vol. 11. №11: e0165826.; Cowley D., Donato С.М., Roczo-Farkas S., Kirkwood C.D. Emergence of a novel equine-like G3P[8] inter-genogroup reassortant rotavirus strain associated with gastroenteritis in Australian children // J Gen Virol. - 2016. Vol. 97. №2. - P. 403-410; Hoa-Tran T.N., Nakagomi T., Vu H.M. et al. Abrupt emergence and predominance in Vietnam of rotavirus A strains possessing a bovine-like G8 on a DS-1-like background // Arch Virol. - 2016. Vol. 161. №2. - P. 479-82; Arana A., Montes M., Jere K.C., Alkorta M., Iturriza-Gomara M., Cilia G. Emergence and spread of G3P[8] rotaviruses possessing an equine-like VP7 and a DS-1-like genetic backbone in the Basque Country (North of Spain), 2015 // Infect Genet Evol. - 2016. №44. - P. 137-144; Kamiya H., Tacharoenmuang R., Ide T. et al. Characterization of an Unusual DS-1-Like G8P[8] rotavirus strain from Japan in 2017: Evolution of emerging DS-1-Like G8P[8] strains through reassortment // Jpn. J. Infect Dis. - 2019. Vol. 72. №4. - P. 256-260]. Преимуществом полногеномной классификации является также возможность выявить и охарактеризовать реассортантные варианты ротавируса, имеющие смешанный набор генов 1 и 2 геногрупп.
В настоящее время для косвенной дифференциации первой и второй геногрупп ротавирусов с помощью мультиплексной ПЦР используют методы, базирующиеся на определении генотипа одного (I-генотип, ген VP6) или двух (I- и Е-генотип, гены VP6 и NSP4) генов в дополнение к G- и Р-генотипам [Бахтояров Г.Н., Киселев И.С., Зверев В.В. и др. Оценка эффективности применения мультиплексной ПЦР в режиме реального времени для генотипирования ротавирусов группы А // Журн. микробиол. - 2014. - №4. - С. 43-49.; Сашина Т.А., Морозова О.В., Епифанова Н.В., Новикова Н.А. Идентификация I- и Е- генотипов ротавируса А с использованием мультиплексной ПЦР // Вопросы вирусологии. - 2019. - Т. 64. - №3. - С. 140-144]. Это позволяет частично охарактеризовать принадлежность штамма к геногруппе на основе двух генов или выявить реассортантые штаммы, если обмен сегментами затронул ген VP6 или NSP4. Однако данная методика не позволяет получить полную информацию о геногрупповой принадлежности штамма или выявить реассортантный штамм, если обмен сегментами затронул другие гены (VP1, VP2, VP3, NSP1, NSP2, NSP3, NSP5/6).
Секвенирование полных нуклеотидных последовательностей всех сегментов генома или их фрагментов признается надежным инструментом для определения полного генотипа ротавирусов. Существует несколько подходов к получению нуклеотидных последовательностей всех сегментов генома ротавирусов: амплификация множества перекрывающихся фрагментов с использованием набора праймеров, фланкирующих перекрывающиеся участки генов длиной не более 600 н.п., и дальнейшее секвенирование по методу Ф. Сэнгера [McDonald S.M., Matthijnssens J., McAllen J.K., Hine E., Overton L., Wang S., Lemey P., Zeller M., Van Ranst M., Spiro D.J., Patton J.T. Evolutionary dynamics of human rotaviruses: balancing reassortment with preferred genome constellations // PLoS Pathog. - 2009. Vol. 5. №10: e1000634]; праймер-опосредованная прогулка (primer walking) [Matthijnssens J., Ciarlet M., Heiman E., Arijs I., Delbeke T., McDonald S.M., Palombo E.A., Iturriza-Gomara M., Maes P., Patton J.T., Rahman M., Van Ranst M. Full genome-based classification of rotaviruses reveals a common origin between human Wa-Like and porcine rotavirus strains and human DS-1-like and bovine rotavirus strains // J Virol. - 2008. Vol. 82. №7. - P. 3204-3219]; амплификация полных последовательностей всех генов с помощью концевых праймеров с дальнейшим анализом на различных NGS-платформах [Faizuloev Е., Mintaev R., Petrusha О., Marova A., Smirnova D., Ammour Y., Meskina E., Sergeev O., Zhavoronok S., Karaulov A., Svitich O., Zverev V. New approach of genetic characterization of group A rotaviruses by the nanopore sequencing method // J. Virol. Methods. - 2021. Vol. 292], создание библиотек для секвенирования на NGS-платформах, осуществляющих короткие чтения, напрямую из РНК ротавируса, выделенной из нативного материала [Shimada S. et al. Use of S1 nuclease in deep sequencing for detection of double-stranded RNA viruses // J Vet Med Sci. - 2015. Vol. 77. №9. - P. 1163-1166]. Сложность использования разных методов секвенирования состоит в том, что они требуют проведения дополнительных этапов пробоподготовки, наличия сложного оборудования, и, таким образом, не подходит для рутинного применения.
В качестве аналога рассматривается методика, предусматривающая ПЦР-амплификацию фрагментов кДНК генома ротавирусов, соответствующих типоспецифической области каждого гена, с применением общих для всех генотипов одного сегмента олигонуклеотидных праймеров, фланкирующих участок длиной около 700 н.п., и определением нуклеотидных последовательностей фрагментов путем секвенирования по методу Ф. Сэнгера и анализом путем их сравнения с имеющимися в базе данных GenBank [Sashina Т.А., Morozova О.V., Epifanova N.V., Novikova N.A. Genotype constellations of the rotavirus A strains circulating in Nizhny Novgorod, Russia, 2017-2018 // Infect Genet Evol. - 2020 Vol. 85].
Задача заявляемого изобретения - разработка способа определения полного генотипа ротавирусов первой и второй геногрупп методом мультиплексной ПЦР.
Технический результат от реализации заявляемого изобретения - способ определения полного генотипа ротавирусов первой и второй геногрупп, обеспечивающий дифференциальную идентификацию ротавирусов первой и второй геногрупп, выявление и характеристику межгеногрупповых реассортантов на основе всех сегментов, включая наименее изученные.
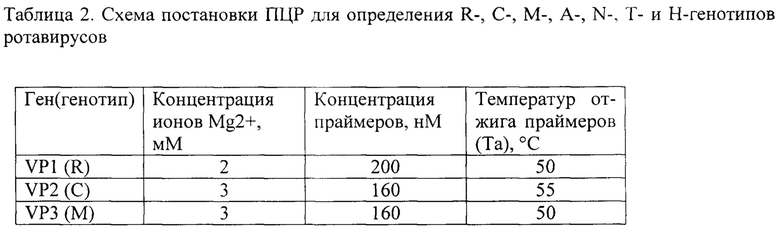
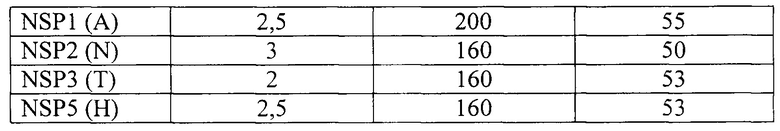
Указанный технический результат достигается определением генотипа каждого из 11 сегментов генома ротавирусов в серии мультиплексных ПЦР с использованием общего праймера (прямого или обратного), комплементарного участку последовательности гена, консервативному среди ротавирусов обеих геногрупп, и двух генотип-специфичных праймеров (прямых или обратных), направленных на вариабельные области.
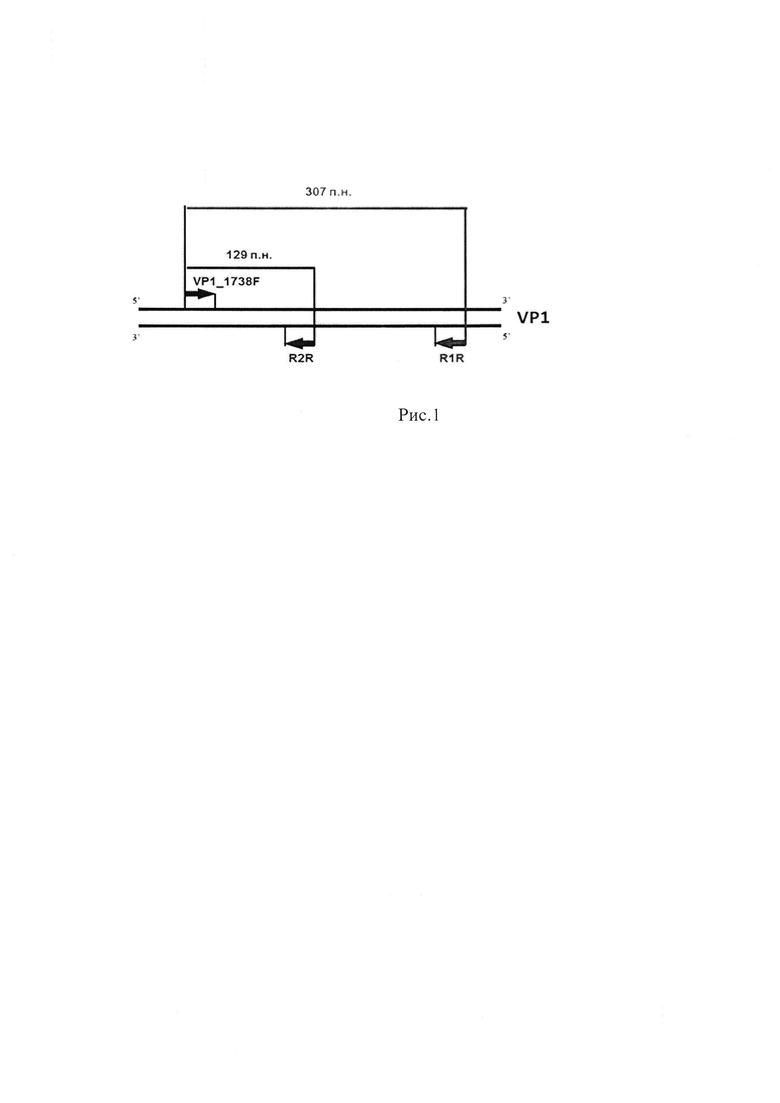
Сущность способа поясняется чертежом.
На рис. 1 представлена Схема посадки праймеров на последовательность гена VP1 для определения R-генотипа.
Способ осуществляется следующим образом.
Из первичного ротавирус-содержащего материала выделяют РНК, которую используют в качестве матрицы для получения кДНК в реакции обратной транскрипции согласно общепринятым методикам. Далее осуществляют постановку серии мультиплексных ПЦР с использованием олигонуклеотидных праймеров SEQ ID NO 1-21 (табл. 1), на основе которых для каждого гена готовят реакционную смесь в объеме 15 мкл следующего состава: ген VP1: SEQ ID NO 1, SEQ ID NO 2, SEQ ID NO 3; ген VP2: SEQ ID NO 4, SEQ ID NO 5, SEQ ID NO 6; ген VP3: SEQ ID NO 7, SEQ ID NO 8, SEQ ID NO 9; ген NSP1: SEQ ID NO 10, SEQ ID NO 11, SEQ ID NO 12; ген NSP2: SEQ ID NO 13, SEQ ID NO 14, SEQ ID NO 15; ген NSP3: SEQ ID NO 16, SEQ ID NO 17, SEQ ID NO 18; ген NSP5/6: SEQ ID NO 19, SEQ ID NO 20, SEQ ID NO 21. В каждую пробирку вносят 0,1 мкл Taq-полимеразы (5 ед/мкл), 0,9 мкл смеси дНТФ (по 2 мМ каждого), 7,8 мкл деионизованной воды, свободной от нуклеаз. Объемы реагентов приведены из расчета на одну пробу. Объемы праймеров и ПЦР-буфера, содержащего ионы магния, берут в реакцию в соответствии с табл. 2. К смеси добавляют 3 мкл кДНК. Амплификацию проводят по программе: 95°С - 1 мин, (95°С - 10 с; Та - 10 с; 75°С - 10 с) × 42 цикла, 75°С - 5 мин.

Примечание: N - любой из А, Т, G или С, R - А или G, Y - С или Т, S - G или С, W - Т или А.
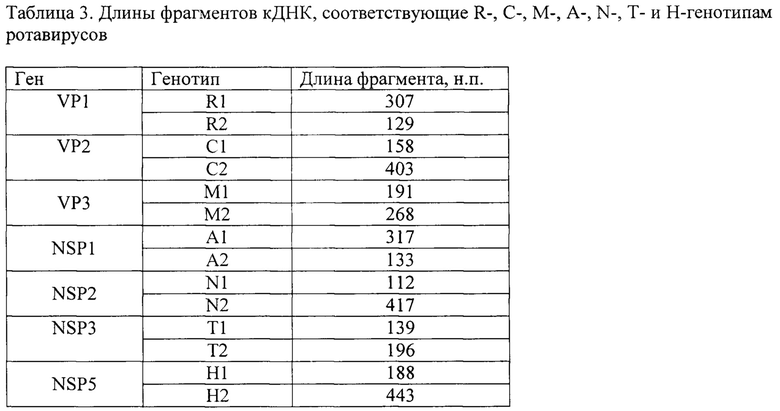
Продукты ПЦР-амплификации визуализируют с помощью горизонтального электрофореза в 1,5% агарозном геле, содержащем бромид этидия. Генотип каждого сегмента определяют по длине получившегося фрагмента кДНК в соответствии с маркером длин ДНК (табл. 3).
Сущность способа поясняется примерами.
Пример 1. Определение полного генотипа ротавирусов первой геногруппы
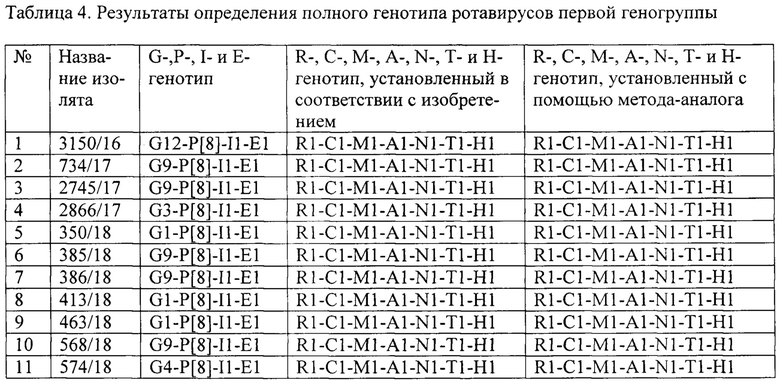
В анализ взяли 11 образцов ротавируса G-, Р-, I- и Е-генотипов, которые были определены методом ПЦР и/или секвенирования по Сэнгеру. Для подтверждения принадлежности ротавирусов к первой геногруппе отобрали характерные для нее ротавирусы генотипов G1-P[8]-I1-E1, G3-P[8]-I1-E1, G4-P[8]-I1-E1, G9-P[8]-I1-E1 и G12-P[8]-I1-E1. Для исследования использовали ротавирус-содержащие фекальные экстракты. РНК ротавирусов выделяли и далее использовали в качестве матрицы для получения кДНК в реакции обратной транскрипции при помощи коммерческих наборов «РИБО-преп» и «РЕВЕРТА-L» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия) в соответствии с инструкциями производителя. Мультиплексную ПЦР на матрице кДНК ротавирусов проводили в отдельной пробирке для каждого анализируемого сегмента. Для постановки ПЦР готовили смесь в объеме 15 мкл, содержащую 0,1 мкл Taq-полимеразы (5 ед/мкл), 0,9 мкл смеси дНТФ (по 2 мМ каждого), 7,8 мкл деионизованной воды, 3 мкл кДНК. Комбинацию праймеров, их количество и объем ПЦР-буфера брали в соответствии с табл. 2. Амплификацию проводили по программе: 95°С - 1 мин, (95°С - 10 с; Та - 10 с; 75°С - 10 с) × 42 цикла, 75°С - 5 мин, где Та для каждого сегмента выставлялась в соответствии с табл. 2. Продукты ПЦР-амплификации визуализировали с помощью горизонтального электрофореза в 1,5% агарозном геле, содержащем бромид этидия. Генотип каждого сегмента определяли по длине получившегося фрагмента кДНК в соответствии с маркером длин ДНК (табл. 3). Дополнительно определили R-, С-, М-, А-, N-, Т- и Н-генотипов методом секвенирования типоспецифических областей анализируемых сегментов по Сенгеру. Результаты представлены в табл. 4.
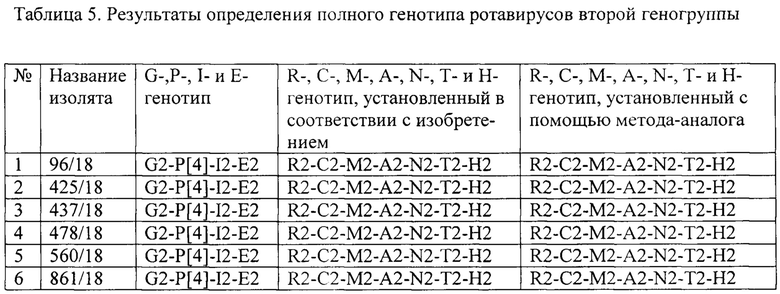
Пример 2. Определение полного генотипа ротавирусов второй геногруппы
Для подтверждения принадлежности ротавирусов ко второй геногруппе были отобраны 6 ротавирусов генотипа G2-P[4]-I2-E2, которые предварительно охарактеризовали методом ПЦР и/или секвенирвоания по Сэнгеру. Провели мультиплексную ПЦР-амплификацию фрагментов кДНК способом в соответствии с изобретением. Дополнительно определили R-, С-, М-, А-, N-, Т- и Н-генотипов методом секвенирования типоспецифических областей анализируемых сегментов по Сэнгеру. Результаты представлены в табл. 5.
Пример 3. Характеристика штаммов, являющихся реассортантами между первой и второй геногруппами.
Для исследования подобрали 11 образцов, содержавших ротавирусы с нетипичным сочетанием генотипов, которое они получили в результате межгеногрупповой реассортации. Провели определение полного генотипа способом в соответствии с изобретением. Дополнительно определили R-, С-, М-, А-, N-, Т- и Н-генотипов методом секвенирования типоспецифических областей анализируемых сегментов по Сэнгеру. Результаты представлены в табл. 6.
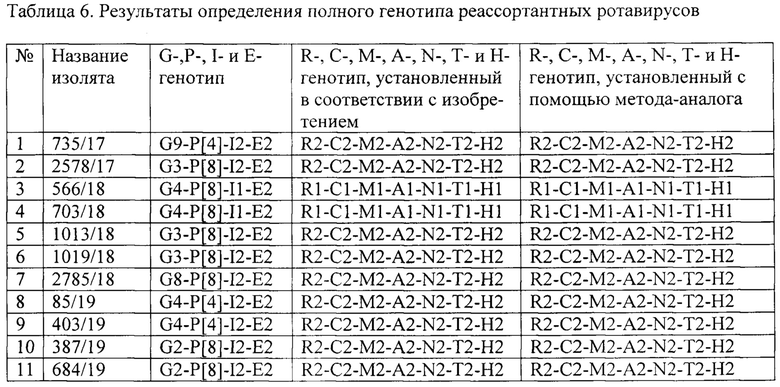
Как видно из табл. 6, два штамма генотипа G4P[8] оказались монореассортантными и несли один ген второй геногруппы (NSP4) в сочетании с остальными генами первой геногруппы. Пять штаммов также являлись монореассортантными и имели один ген (VP7 или VP4) первой геногруппы в сочетании с остальными генами второй геногруппы. Четыре штамма были двойными реассортантами и характеризовались наличием двух генов первой геногруппы (VP7 и VP4) и девяти генов второй геногруппы, что подтвердили с помощью секвенирования.
Проведенные исследования подтвердили, что использование заявленного изобретения позволяет достоверно за счет подобранных типоспецифичных праймеров для мультипраймерной ПЦР не только дифференциально идентифицировать ротавирусы первой и второй геногруппы, но и выявлять межгеногрупповые реассортанты даже в том случае, когда реассортация затронула наименее изученные сегменты, с целью мониторинга циркуляции штаммов ротавирусов, изучения их распространенности и вариабельности.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="СПОСОБ ОПРЕДЕЛЕНИЯ
ПОЛНОГО ГЕНОТИПА РОТАВИРУСОВ.xml" softwareName="WIPO Sequence"
softwareVersion="2.3.0" productionDate="2024-02-21">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText></ApplicationNumberText>
<FilingDate></FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>1</ApplicantFileReference>
<ApplicantName languageCode="ru">Федеральное бюджетное учреждение
науки «Нижегородский научно-исследовательский институт эпидемиологии
и микробиологии им. академика И.Н. Блохиной» Федеральной службы по
надзору в сфере защиты прав потребителей и благополучия человека
</ApplicantName>
<ApplicantNameLatin>Academician I.N. Blokhina Nizhny Novgorod
Scientific Research Institute of Epidemiology and
Microbiology</ApplicantNameLatin>
<InventorName languageCode="ru">Татьяна Александровна
Сашина</InventorName>
<InventorNameLatin>Tatiana A. Sahina</InventorNameLatin>
<InventionTitle languageCode="ru">СПОСОБ ОПРЕДЕЛЕНИЯ ПОЛНОГО
ГЕНОТИПА РОТАВИРУСОВ ПЕРВОЙ И ВТОРОЙ ГЕНОГРУПП МЕТОДОМ
МУЛЬТИПЛЕКСНОЙ ПЦР</InventionTitle>
<SequenceTotalQuantity>21</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtwcaaataccwgatggwaatgt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caataccgactgtagataccaat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agaaatacgtgataaaacyg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggtcatatctccacagtgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccttttgatttyagagcgtca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtagcattggttggagcacttc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggtcacatcrtgacyagtgtg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q16">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcacatcacgtttggacaatcac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q18">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctgatgaggtgtaygtggc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q20">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctttttttatgaaaagtcttgtg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q22">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cattaaatcatttttcgtcacttc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q24">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agagtagaaggtctccaaaca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="13">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q26">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtatthccttgtttcattgarga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="14">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q28">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>catataagaataatgctaagagtgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="15">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q30">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttcaaargagattgaaacractgcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="16">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q32">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caaaaratgagagtvctkaatgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="17">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q34">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>catcaacatatgaatcatcaacc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="18">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q36">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtctttcaagttgatcatatctgga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="19">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q38">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggtcacaaaacgggagtgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="20">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q40">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttctgattctgaagattatgttttgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="21">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q42">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttgatgctacaatggattcytc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| СПОСОБ ГЕНОТИПИРОВАНИЯ ИЗОЛЯТОВ NEISSERIA GONORRHOEAE НА БИОЛОГИЧЕСКОМ МИКРОЧИПЕ | 2023 |
|
RU2816767C1 |
| Способ мультиплексной идентификации 32 генетических маркеров льна | 2022 |
|
RU2804939C1 |
| Иммунобиологическое средство на основе вирусоподобных частиц для индукции специфического иммунитета против инфекции, вызываемой ротавирусом А человека | 2022 |
|
RU2795055C1 |
| СПОСОБ ОПРЕДЕЛЕНИЯ СУБЛИНИЙ ВОЗБУДИТЕЛЯ ТУБЕРКУЛЕЗА ЛИНИИ L2 Beijing НА БИОЛОГИЧЕСКИХ МИКРОЧИПАХ | 2022 |
|
RU2790296C1 |
| НАБОР ОЛИГОНУКЛЕОТИДОВ И СПОСОБ ОБОГАЩЕНИЯ ГЕНОМНОЙ КДНК ВИРУСА КРЫМСКОЙ-КОНГО ГЕМОРРАГИЧЕСКОЙ ЛИХОРАДКИ ГЕНОТИПА ЕВРОПА-1 (V) МЕТОДОМ ПОЛИМЕРАЗНОЙ ЦЕПНОЙ РЕАКЦИИ | 2023 |
|
RU2839313C1 |
| Способ мультиплексной идентификации 52 генетических маркеров хозяйственно ценных признаков льна | 2023 |
|
RU2826718C1 |
| НАБОР ОЛИГОНУКЛЕОТИДНЫХ ПРАЙМЕРОВ ДЛЯ ГЕНОТИПИРОВАНИЯ ГРИБОВ ВИДОВОГО КОМПЛЕКСА COLLETOTRICHUM ACUTATUM | 2022 |
|
RU2807352C1 |
| Способ сполиготипирования микобактерий туберкулезного комплекса с использованием ДНК-амплификации в иммобилизованной фазе и биологический микрочип для его осуществления | 2023 |
|
RU2807998C1 |
| Вирусоподобные химерные частицы для индукции специфического иммунитета против вируса тяжелого острого респираторного синдрома SARS-CoV-2, содержащие белки коронавируса и ротавируса | 2022 |
|
RU2779810C1 |
| Нуклеотидная последовательность, кодирующая фермент литиказу, и панель олигонуклеотидов для получения синтетической нуклеотидной последовательности гена литиказы | 2023 |
|
RU2826150C1 |
Изобретение относится к области молекулярной биологии, вирусологии и молекулярно-генетической диагностики. Описан способ генотип-специфичной амплификации кДНК ротавирусов методом мультиплексной полимеразной цепной реакции. В способе, включающем выделение вирусной РНК, получение кДНК, которую используют в качестве матрицы при постановке ПЦР с электрофоретической визуализацией продуктов амплификации в агарозном геле, проводят серию ПЦР с использованием олигонуклеотидов SEQ ID NO 1-21 для формирования реакционных смесей, в которых олигонуклеотиды SEQ ID NO 1-21 распределены по реакционным смесям следующим образом для выявления: гена VP1: SEQ ID NO 1, SEQ ID NO 2, SEQ ID NO 3; гена VP2: SEQ ID NO 4, SEQ ID NO 5, SEQ ID NO 6; гена VP3: SEQ ID NO 7, SEQ ID NO 8, SEQ ID NO 9; гена NSP1: SEQ ID NO 10, SEQ ID NO 11, SEQ ID NO 12; гена NSP2: SEQ ID NO 13, SEQ ID NO 14, SEQ ID NO 15; гена NSP3: SEQ ID NO 16, SEQ ID NO 17, SEQ ID NO 18; гена NSP5/6: SEQ ID NO 19, SEQ ID NO 20, SEQ ID NO 21. Изобретение за счет подобранных праймеров для мультиплексной ПЦР позволяет достоверно не только дифференциально идентифицировать ротавирусы первой и второй геногруппы, но и выявлять межгеногрупповые реассортанты в случае, когда реассортация затронула наименее изученные сегменты, с целью мониторинга циркуляции ротавирусов и изучения их распространенности и вариабельности. 4 з.п. ф-лы, 1 ил., 6 табл., 3 пр.
1. Способ определения генотипа ротавирусов первой и второй геногрупп и ре-ассортантов между ними методом мультиплексной ГИДР, включающий выделение вирусной РНК, получение кДНК, которую используют в качестве матрицы при постановке ПЦР с электрофоретической визуализацией продуктов амплификации в агарозном геле, отличающийся тем, что в серии ПЦР используют олигонуклеотиды SEQ ID NO 1-21 для формирования реакционных смесей, в которых олигонуклеотиды SEQ ID NO 1-21 распределены по реакционным смесям следующим образом для выявления: гена VP1: SEQ ID NO 1, SEQ ID NO 2, SEQ ID NO 3; гена VP2: SEQ ID NO 4, SEQ ID NO 5, SEQ ID NO 6; гена VP3: SEQ ID NO 7, SEQ ID NO 8, SEQ ID NO 9; гена NSP1: SEQ ID NO 10, SEQ ID NO 11, SEQ ID NO 12; гена NSP2: SEQ ID NO 13, SEQ ID NO 14, SEQ ID NO 15; гена NSP3: SEQ ID NO 16, SEQ ID NO 17, SEQ ID NO 18; гена NSP5/6: SEQ ID NO 19, SEQ ID NO 20, SEQ ID NO 21, причем длина фрагмента кДНК соответствует: 307 н.п. - генотипу R1 гена VP1, 129 н.п. - генотипу R2 гена VP1, 158 н.п. - генотипу С1 гена VP2, 403 н.п. – генотипу С2 гена VP2, 191 н.п.- генотипу M1 гена VP3, 268 н.п.- генотипу М2 гена VP3, 317 н.п. - генотипу А1 гена NSP1, 133 н.п. - генотипу А2 тепа NSP1, 112 н.п. - генотипу N1 гена NSP2, 417 н.п. - генотипу N2 тепа NSP2, 139 н.п. - генотипу Т1 гена NSP3, 196 н.п. - генотипу 12 гена NSP3, 188 н.п. - генотипу H1 гена NSP5, 443 н.п. - генотипу Т2 гена NSP3.
2. Способ по п. 1, отличающийся тем, что для каждого гена готовят отдельную реакционную смесь в объеме 15 мкл.
3. Способ по пп. 1 и 2, отличающийся тем, что в каждую пробирку вносят 0,1 мкл Taq-полимеразы 5 ед/мкл, 0,9 мкл смеси дНТФ по 2 мМ каждого, 7,8 мкл деионизованной воды, свободной от нуклеаз, с добавлением 3 мкл кДНК, причем праймеры нМ и Mg2+-содержащий ПЦР-буфер мМ берут в реакцию в концентрации: ген VP1 генотип R - 200,0 и 2,0; ген VP2 генотип С - 160,0 и 3,0; ген VP3 генотип М - 160,0 и 3,0; ген NSP1 генотип А - 200,0 и 2,5; ген NSP2 генотип N - 160,0 и 3,0; ген NSP3 генотип Т - 160,0 и 2,0; ген NSP5 генотип Н - 160,0 и 2,5.
4. Способ по пп. 1-3, отличающийся тем, что амплификацию проводят по программе: 95°С - 1 мин, (95°С - 10 с; Та - 10 с; 75°С - 10 с) × 42 цикла, 75°С - 5 мин.
5. Способ по п. 4, отличающийся тем, что отжиг праймеров осуществляют при температуре °С для генов VP1 R, VP3 М, NSP2 - 50; NSP3 Т, NSP5R - 53; VP2, NSP1 А - 55.
| О.В | |||
| МОРОЗОВА, Генотиповое разнообразие и филодинамика циркулирующих на территории России ротавирусов, сравнение с вакцинными штаммами, Диссертация на соискание ученой степени кандидата биологических наук, Москва, 2021, с | |||
| Джино-прядильная машина | 1922 |
|
SU173A1 |
Авторы
Даты
2025-01-14—Публикация
2024-03-06—Подача