Изобретение относится к биотехнологии, к ветеринарной вирусологии и молекулярной биологии и представляет способ детекции ДНК вируса африканской чумы свиней генотипа II. Изобретение может быть использовано в ветеринарии.
Вирус африканской чумы свиней (АЧС) вызывает тяжелое, острозаразное заболевание со смертельным исходом как у домашних, так и у диких свиней. Вирус АЧС приводит к огромным экономическим потерям, поэтому быстрый и доступный тест для определения АЧС непосредственно на ферме или в ближайшем ветеринарном пункте является значимым для борьбы с эпизоотией.
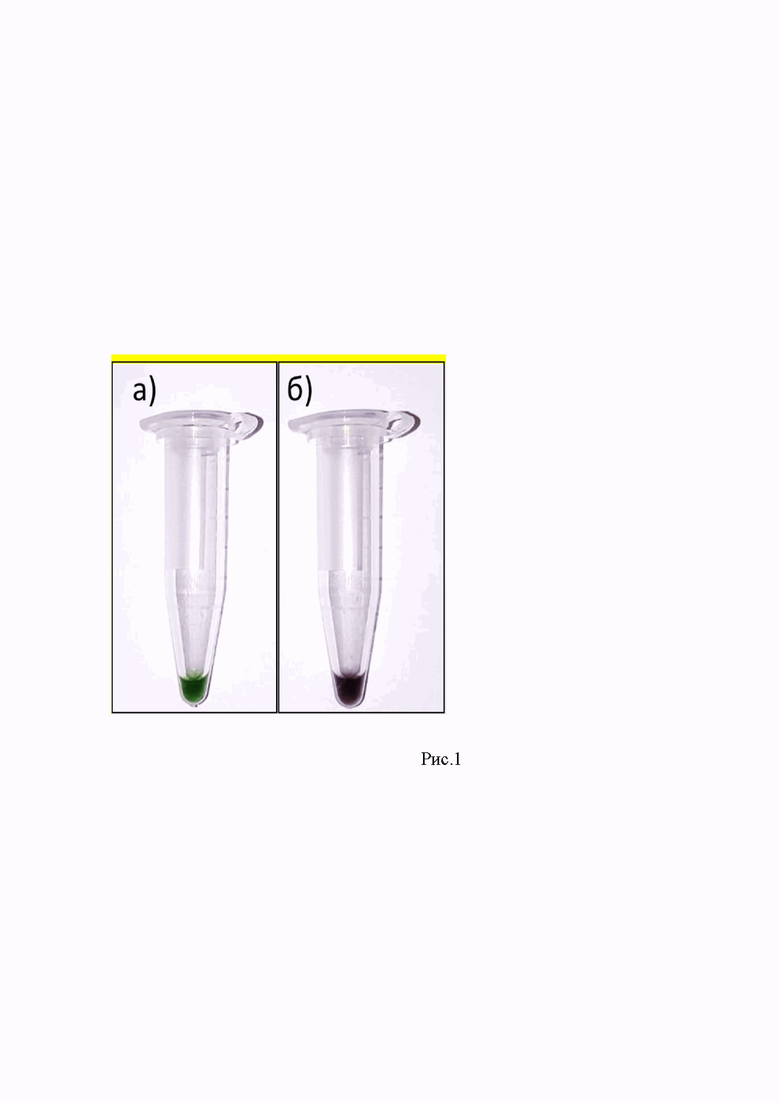
Предлагаемый способ детекции осуществляется с помощью специфических олигонуклеотидов (праймеров) путем изотермической петлевой амплификации (LAMP) и колориметрической детекции результатов амплификации с помощью pH-чувствительных красителей. На рис. 1 демонстрируется пример изменения цвета реакционной смеси при а) обнаружении ДНК вируса АЧС в пробе б) отсутствии ДНК вируса АЧС в пробе.
Уровень техники
На настоящий момент в России вирус АЧС определяется методом ПЦР в реальном времени. На основе этого метода было разработано несколько систем для обнаружения вируса АЧС: RU2645263C1, RU2360971C1, RU2606253C1 [1-3]. Созданный нами способ детекции на основе LAMP более доступен по сравнению с ПЦР в реальном времени. Для проведения детекции методом LAMP требуется наличие термостата на 60°С, в отличие от приведенных выше патентов, требующих сложных приборов для осуществления детекции в режиме ПЦР в реальном времени, что является преимуществом нашего способа и позволяет использовать его на любом ветеринарном пункте или на ферме и осуществлять быструю диагностику при подозрении на инфекцию.
Петлевая изотермическая амплификация (LAMP) - метод изотермической амплификации ДНК, в котором используется полимераза с вытесняющей активностью и, как минимум, 2 пары праймеров: внутренние и внешние. Каждый из внутренних праймеров имеет последовательность, комплементарную одной цепи участка амплификации на 3'-конце и идентичную внутреннему участку той же цепи на 5'-конце, в результате чего в ходе реакции образуются петлевые структуры, число которых увеличивается с каждым циклом реакции [4]. Поскольку используются четыре специфических праймера, которые распознают шесть различных последовательностей ДНК-мишени, LAMP амплифицирует ДНК с высокой специфичностью.
Сравнение аналитической чувствительности между обычными ПЦР, Nested polymerase chain reaction, ПЦР в реальном времени и LAMP показало, что LAMP превосходит другие методы по таким параметрам, как предел обнаружения и время амплификации [5]. Кроме того, LAMP менее чувствительна к присутствию в реакционной смеси различных биологических примесей, чем ПЦР [6], что упрощает пробоподготовку образцов.
Ключевым моментом в разработке эффективного способа выявления патогенов является подбор специфических олигонуклеотидов, обеспечивающих прохождение реакции. Геном вируса АЧС характеризуется значительной изменчивостью. Олигонуклеотиды, раскрытые в настоящем изобретении, специфичны для наиболее распространенных на территории РФ штаммов вируса АЧС.
Наиболее близким аналогом предлагаемого технического решения является изобретение № RU2710065C1 [7], раскрывающее олигонуклеотидные праймеры для выявления ДНК вируса африканской чумы свиней, подобранные для обнаружения гена вируса р22, а также способ выявления ДНК вируса африканской чумы свиней и с помощью петлевой изотермической амплификации LAMP. Для регистрации результатов используется интеркалирующий флуоресцентный краситель Sybr Green или турбидиметрия.
Предлагаемое техническое решение отличается геном-мишенью для детекции вируса и составом нуклеотидных праймеров: были разработаны праймеры, специфические к консервативному участку гену капсидного белка p72.
Отличительной чертой заявленного способа является возможность визуальной детекции по изменению цвета реакционной смеси, основанное на использовании индикатора рH, дополненного красителем, создающим более контрастный цветовой переход. Красители добавляются перед реакцией, что снижает вероятность загрязнения амликонами при открывании пробирки. В отличие от флуорохромов, которые также могут использоваться для детекции, указанные индикаторы не подвержены фотообесцвечиванию.
В заявленном техническом решении были оптимизированы концентрации компонентов буферного раствора, обеспечивающие активность Bst полимеразы и при этом позволяющие зафиксировать изменение pH. Для обнаружения изменения pH в составе смеси присутствуют индикатор и краситель, обеспечивающие контрастный визуальный переход при положительной реакции. Существенными признаками, отличающими заявляемое техническое решение от аналогов, являются:
- нуклеотидные последовательности праймеров
- оптимизированный состав амплификационной смеси
- визуальная детекция результата реакции с использованием pH индикатора и красителя
ПРИМЕР 1
Реакционная смесь приготовлена при охлаждении на льду. Состав реакционной смеси был следующим:
1,5 ммоль/л Tris-HCl, pH=8,8
10 ммоль/л (NH4)2SO4
10 ммоль/л KCl
6 ммоль/л MgSO4
0,4 ммоль/л ATP,
0,4 ммоль/л CTP,
0,4 ммоль/л GTP,
0,4 ммоль/л TTP,
0,1 % Triton X-100
0,2 мкмоль/л олигонуклеотида ASF-F3
0,2 мкмоль/л олигонуклеотида ASF-B3
1,6 мкмоль/л олигонуклеотида ASF-FIP
1,6 мкмоль/л олигонуклеотида ASF-BIP
50 мкмоль/л метиленового синего
50 мкмоль/л фенолового красного
1 мкл Bst полимеразы (16 е.а.)
деионизированной воды, свободной от нуклеаз - до 24 мкл
1 мкл анализируемого образца, содержащего ДНК вируса АЧС добавляли в 24 мкл реакционной смеси. В качестве отрицательного контроля вместо раствора анализируемого образца использовали 1 мкл деионизованной воды. Реакцию проводили путем нагревания в термостате при 60°С в течении 50 минут. В результате проведения реакции в положительной пробе произошло изменение цвета реакционной смеси с фиолетового на зеленый, в отрицательном контроле цвет реакционной смеси не изменился рис.1.
ПРИМЕР 2
Реакционная смесь в объеме 25 мкл была приготовлена при охлаждении на льду. Состав реакционной смеси был следующий:
3 ммоль/л Tris-HCl, pH=8,8
10 ммоль/л (NH4)2SO4
10 ммоль/л KCl
6 ммоль/л MgSO4
0,4 ммоль/л ATP,
0,4 ммоль/л CTP,
0,4 ммоль/л GTP,
0,4 ммоль/л TTP,
0,1 % Triton X-100
0,2 мкмоль/л олигонуклеотида ASF-F3
0,2 мкмоль/л олигонуклеотида ASF-B3
1,6 мкмоль/л олигонуклеотида ASF-FIP
1,6 мкмоль/л олигонуклеотида ASF-BIP
50 мкмоль/л метиленового синего
50 мкмоль/л фенолового красного
1 мкл Bst полимеразы (16 е.а. )
деионизированной воды, свободной от нуклеаз - до 24 мкл
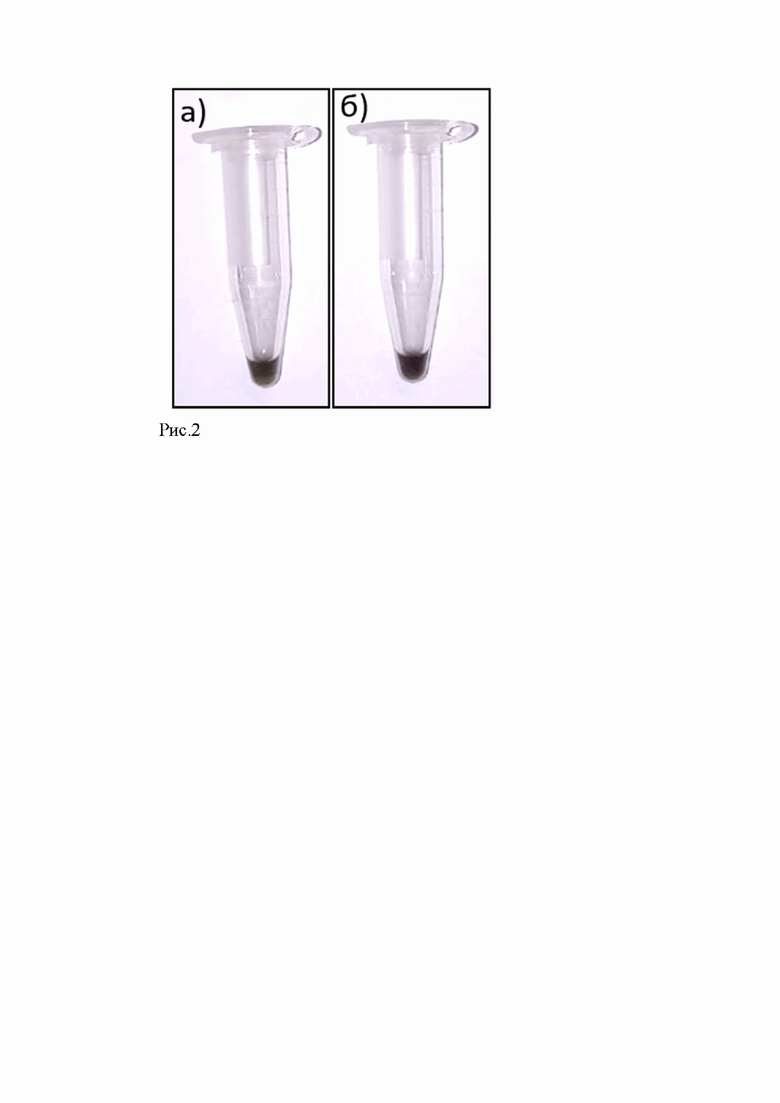
1 мкл анализируемого образца, содержащего ДНК вируса АЧС добавляли в 24 мкл реакционной смеси. В качестве отрицательного контроля вместо раствора анализируемого образца использовали 1 мкл деионизованной воды. Реакцию проводили путем нагревания в термостате при 60°С в течении 50 минут. В результате проведения реакции в пробе с контрольной ДНК и в отрицательном контроле цвет реакционной смеси не изменился рис.2. ПРИМЕР 2 отличался от ПРИМЕРА 1 буферной емкостью раствора, изменением которой удалось достигнуть оптимального состава реакционной смеси, обеспечивающей прохождение реакции и возможность регистрации результатов.
Источники информации
1. Потапова А. Ю., Сердюк И. Ю., Джаилиди Г. А., Черных О. Ю., Кривонос Р. А., Кощаев А. Г., Лысенко А. А., Шевченко А. А.; ФГБОУ ВО "Кубанский государственный аграрный университет имени И.Т. Трубилина", ООО "Инновационные диагностические системы". Тест-система для обнаружения ДНК вируса африканской чумы свиней с помощью полимеразной цепной реакции в режиме реального времени 2017. Патент РФ № RU 2645263 C1 Дата заявки: 05.04.2017
2. Калабеков И.М., Жигалева О.Н., Власова Н.Н., Цыбанов С.Ж., Колбасов Д.В.; ГНУ ВНИИ ветеринарной вирусологии и микробиологии. Способ и тест-система для обнаружения ДНК вируса африканской чумы свиней с помощью специфических олигонуклеотидных праймеров в полимеразной цепной реакции. Патент РФ № RU2360971C1. Дата заявки 23.11.2007
3. Кудряшов, Д. А., Бурмакина, Г. С., Титов, И. А., Цыбанов, С. Ж., Малоголовкин, А. С.; Всероссийский научно-исследовательский институт ветеринарной вирусологии и микробиологии Россельхозакадемии. Олигонуклеотидные праймеры и флюоресцентный зонд с внутренним гасителем, комплементарные участку гена Р30 (CP204L) вируса африканской чумы свиней, для использования в полимеразной цепной реакции в режиме реального времени. Патент РФ № RU 2606253 C1. Дата заявки 15.02.2016
4. Notomi T., Mori Y., Tomita N., Kanda H. Loop-mediated isothermal amplification (LAMP): principle, features, and future prospects // J. Microbiol. 2015. Vol. 53, № 1. P. 1-5.
5. Foo P.C., Nurul Najian A.B., Muhamad N.A., Ahamad M., Mohamed M., Yean Yean C., Lim B.H. Loop-mediated isothermal amplification (LAMP) reaction as viable PCR substitute for diagnostic applications: a comparative analysis study of LAMP, conventional PCR, nested PCR (nPCR) and real-time PCR (qPCR) based on Entamoeba histolytica DNA derived from // BMC Biotechnol. 2020. Vol. 20, № 1. P. 34.
6. Kaneko H., Kawana T., Fukushima E., Suzutani T. Tolerance of loop-mediated isothermal amplification to a culture medium and biological substances // J. Biochem. Biophys. Methods. 2007. Vol. 70, № 3. P. 499-501
7. Елсукова А. А., Власова Н. Н., Иголкин А. С.; ФГБУ "ВНИИЗЖ”. Синтетические олигонуклеотидные праймеры и способ выявления ДНК вируса АЧС методом петлевой изотермической амплификации. Патент РФ № RU2710065C1. Дата заявки 14.12.2018
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="Способ обнаружения
ДНК вируса африканской чумы свиней с помощью специфических
олигонуклеотидов путем изотермической петлевой амплификации и
колориметрической детекции результатов амплификации.xml"
softwareName="WIPO Sequence" softwareVersion="2.2.0"
productionDate="2023-01-10">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2022134169</ApplicationNumberText>
<FilingDate>2022-12-23</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>https://www.fips.ru/registers-doc-view/fips_s
ervlet?DB=RUPATAP&DocNumber=2022134169&TypeFile=html</Applican
tFileReference>
<ApplicantName languageCode="ru">Федеральное государственное
учреждение «Федеральный исследовательский центр «Фундаментальные
основы биотехнологии» Российской академии наук»</ApplicantName>
<ApplicantNameLatin>Federal Research Centre "Fundamentals of
Biotechnology" of the Russian Academy of
Sciences</ApplicantNameLatin>
<InventorName languageCode="ru">Гончаренко Анна
Владимировна</InventorName>
<InventorNameLatin>Goncharenko Anna Vladimirovna</InventorNameLatin>
<InventionTitle languageCode="ru">Способ обнаружения ДНК вируса
африканской чумы свиней с помощью специфических олигонуклеотидов
путем изотермической петлевой амплификации и колориметрической
детекции результатов амплификации</InventionTitle>
<SequenceTotalQuantity>4</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q13">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgcagaactttgatggaaat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>attatgcagcccactcac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>50</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..50</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q15">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccgttacgtatccgatcacattaccttatcgataagattgataccatga
g</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q16">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcagatatagatgaacatgcgtctgcacgcagagataagctttca</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
Настоящее изобретение относится к области биотехнологии. Описан способ детекции ДНК вируса африканской чумы свиней. Способ включает проведение изотермической петлевой амплификации путем нагревания в термостате в течение 50-70 минут и регистрацию результата анализа по изменению цвета реакционной смеси. Он отличается тем, что в качестве праймеров в реакции изотермической петлевой амплификации используются последовательности олигонуклеотидов ASF-F3: SEQ NO1, ASF-B3: SEQ NO2, ASF-FIP: SEQ NO3, ASF-BIP: SEQ NO4. Технический результат, достигаемый изобретением, заключается в расширении арсенала средств детекции ДНК вируса африканской чумы свиней. 1 з.п. ф-лы, 2 ил., 2 пр.
1. Способ детекции ДНК вируса африканской чумы свиней, включающий проведение изотермической петлевой амплификации путем нагревания в термостате в течение 50-70 минут и регистрацию результата анализа по изменению цвета реакционной смеси, отличающийся тем, что в качестве праймеров в реакции изотермической петлевой амплификации используются последовательности олигонуклеотидов ASF-F3: SEQ NO1, ASF-B3: SEQ NO2, ASF-FIP: SEQ NO3, ASF-BIP: SEQ NO4.
2. Способ детекции ДНК вируса африканской чумы свиней по п.1, отличающийся тем, что реакция проводится в буфере с низкой емкостью в присутствии комбинации индикатора pH фенолового красного и красителя метиленового синего и регистрация результатов реакции осуществляется путем визуальной колориметрической оценки: в отрицательном контроле и в пробах, где отсутствует ДНК вируса АЧС, будет темно-фиолетовое окрашивание, в положительном контроле и в пробах, где присутствует ДНК вируса АЧС, реакционная смесь приобретает зеленый цвет.
| Синтетические олигонуклеотидные праймеры и способ выявления ДНК вируса АЧС методом петлевой изотермической амплификации | 2018 |
|
RU2710065C1 |
| CN 109897889 A, 18.06.2019 | |||
| CN 106521027 A, 22.03.2017. | |||
Авторы
Даты
2023-11-08—Публикация
2022-12-23—Подача