Изобретение относится к разработке набора олигонуклеотидных праймеров для выявления РНК вируса желтой лихорадки в клинических образцах человека (сыворотка крови) и вируссодержащих культуральных жидкостях, с целью идентификации завозных случаев желтой лихорадки на территории Российской Федерации.
Переносимые членистоногими представители семейства Flaviviridae, в том числе и вирус желтой лихорадки (ВЖЛ), представляют большую угрозу для здоровья населения в тропических и субтропических регионах, а в последнее время стали предметом обеспокоенности медицинских работников и в странах с умеренным климатом.
Вирус желтой лихорадки (ВЖЛ) является представителем рода Flavivirus (семейство Flaviviridae), включающий в себя еще порядка 80 видов, и относится ко II группе патогенности [1]. Основным переносчиком ВЖЛ являются комары вида Aedes aegypti, которые обитают как на эндемичных по желтой лихорадке (ЖЛ) территориях (Африка, Южная Америка и др.) [2], так и в не эндемичных (например, юг России, Абхазия и Грузия) [3]. Вирус вызывает тяжелое заболевание, характеризующиеся лихорадкой, рвотой и тошнотой, болью в мышцах и спине, повсеместной желтухой, поражением печени, почек, кровотечениями и другими симптомами. Ежегодно во всем мире происходит примерно 80-200 тыс. случаев ЖЛ с уровнем летальности 20-60%. Истинное количество случаев оценить трудно из-за недостатка данных, т.к. не все люди обращаются в медицинские учреждения после укуса зараженного комара, или из-за отсутствия современных доступных методов диагностики в странах с недостаточно высоким уровнем здравоохранения [4].
По данным ВОЗ за 2016-2023 гг. случаи ЖЛ регистрируются в ряде стран Африки, Центральной и Южной Америки (Ангола, Венесуэла, Гана, Демократическая Республика Конго, Камерун, Кот-д'Ивуар, Нигер, Нигерия, Республика Конго, Центральноафриканская Республика, Чад, Уганда, Бразилия и Республика Суринам), а также встречаются завозные случаи на не эндемичные территории (Китай, Нидерланды, страны Европы) [5-8].
Лабораторные методы выявления возбудителя ЖЛ, как и других геморрагических лихорадок, основаны на обнаружении вируса или его частиц (РНК и специфических антигенов), а также исследовании динамики титра специфических антител, и проводятся с использованием вирусологических, серологических и молекулярно-генетических тестов [9]. Производимые тест-системы для выявления генетического материала ВЖЛ основанные на амплификации нуклеиновых кислот представлены в таблице 1.
Производимые ПЦР тест-системы на 2023 г. [10]
(РЗН 2017/6637 от 26 декабря 2017 года)
(РЗН 2016/4879 от 7 октября 2016 года)
(РЗН 2017/6631)
Rickettsia spp.; Leptospira spp.; Salmonella spp.) (Fast Track Diagnostics, Люксембург)
(ViPrimePLUS, Малайзия)
В настоящее время отечественная диагностика ЖЛ представлена ПЦР тест-системами, время анализа которыми занимает более 3 часов без учета времени, затраченного на доставку проб в лабораторию. Тесты зарубежного производства весьма дороги, а их качество, в ряде случаев, оставляет желать лучшего.
В литературе имеются данные о разработке диагностических наборов, основанных на изотермической амплификации генома ВЖЛ [11-12]. К сожалению, ни один из них в данный момент не производится. Недостатками таких наборов является оценка диагностических характеристик в БОЕ/мл, что не дает прямого соотношения выявления копий нуклеиновых в реакции (или в миллилитре), поскольку нет подтверждение того, что используемая для анализа культуральная жидкость была очищенна от не бляшкообразующих единичных вирусов (например, разрушенных вирусных частиц). Таким образом происходит завышение результатов анализа чувствительности наборов реагентов. Для проверки нашего изобретения был получен плазмидный положительный контроль, который позволил оценить диагностическую чувствительность в копиях/мл.
Наиболее близким аналогом (прототипом) является способ изотермического обнаружения множества патогенов, включающий: получение образца; извлечение нуклеиновой кислоты из образца; смешивание образца с реагентами для амплификации или детекции; помещение образца смеси в устройство термического нагрева на заданный период времени; амплификация экстрагированной нуклеиновой кислоты; и обнаружение как минимум двух патогенов в реальном времени по выделенной нуклеиновой кислоте, при этом этап обнаружения дополнительно включает: приведение образца в контакт с двумя или более наборами праймеров-зондов для петлевой изотермической амплификации (LAMP) в реальном времени (патент США №11473155, МПК C12Q1/6806, опубл. 18.10.2022 г.) [13]. Образцы содержат патогены, содержащие по меньшей мере два или более вирусов, содержащих нуклеиновую кислоту, например, нуклеиновая кислота вируса желтой лихорадки (YFV). Заданный период времени реакции амплификации составляет от около 10 до около 40 минут. Два или более набора праймеров реального времени (RT)-LAMP содержат наборы праймеров (RT)-LAMP, специфичных к нуклеиновой кислоте YFV, содержат от четырех до шести праймеров, состоящих из последовательностей нуклеиновых кислот SEQ ID NO: 176-181.
Недостатками указанного прототипа является то, что заявляемый способ предназначен для получения многочисленных дифференциально-диагностических данных для осуществления целевого терапевтического вмешательства и мониторинга тенденций передачи заболеваний посредством эпидемиологического надзора за распространением многочисленных вирусных инфекций.
Техническим результатом заявляемого изобретения является разработка набора для выявления РНК ВЖЛ методом изотермической амплификации для лабораторной верификации диагноза, выявления ВЖЛ у комаров-переносчиков при изучении возможного распространения возбудителя как в эндемичных, так и в неэндемичных районах.
Указанный технический результат достигается созданием набора олигонуклеотидных праймеров для выявления РНК вируса желтой лихорадки (ВЖЛ) методом изотермической амплификации в режиме реального времени, содержащий 6 праймеров: внешние - F3 и B3, внутренние FIP и BIP, петлевые праймеры - LF и LB, имеющие следующие нуклеотидные последовательности SEQ ID NO: 1-6 соответственно:
- SEQ ID NO: 1. Последовательности олигонуклеотидов видоспецифичных к ВЖЛ (прямой внешний праймер F3 5'→3').
F3: 5'-AAAAGATYACGGCYCACC-3'
- SEQ ID NO: 2. Последовательности олигонуклеотидов видоспецифичных к ВЖЛ (обратный внешний праймер B3 5'→3').
B3: 5'-CAGATGTCACATTTAGGAGC-3'
- SEQ ID NO: 3. Последовательности олигонуклеотидов видоспецифичных к ВЖЛ (прямой внутренний праймер FIP 5'→3').
FIP: 5'-GGCCACCACTCTYTTAACTTTCCTTTTTTGGTTGTGGAAAATGCTGG-3'
- SEQ ID NO: 4. Последовательности олигонуклеотидов видоспецифичных к ВЖЛ (обратный внутренний праймер BIP 5'→3').
BIP: 5'-CCGTTCCCATGATGTTCTGACTTTTTTCCGCAYCAAGGTCACT-3'
- SEQ ID NO: 5. Последовательности олигонуклеотидов видоспецифичных к ВЖЛ (петлевой прямой праймер LF 5'→3').
LF: 5'- ARCAGCCAAGCCTTGTCTTG -3'
- SEQ ID NO: 6. Последовательности олигонуклеотидов видоспецифичных к ВЖЛ (петлевой обратный праймер LB 5'→3').
LB: 5'- TTGGGAATGCTGTTGATGACGG -3'
Разработка набора для выявления РНК ВЖЛ методом изотермической амплификации позволит выявлять РНК вируса менее, чем за 40 минут с детекцией результатов в режиме реального времени.
Технология получения набора олигонуклеотидных праймеров для выявления РНК вируса желтой лихорадки (ВЖЛ) методом изотермической амплификации в режиме реального времени
Дизайн праймеров
Дизайн праймеров, необходимых для получения клонируемых фрагментов гена NS1 осуществляли при помощи программы Primer Explorer V5, используя известные нуклеотидные последовательности геномов ВЖЛ, доступные в базе данных GenBank (http://www.ncbi.nlm.nih.gov/genbank). При конструировании олигонуклеотидных праймеров учитывались следующие требования: отсутствие протяженных участков повторяющихся нуклеотидов, отсутствие комплементарных последовательностей длиной более трех оснований внутри олигонуклеотидов, высокое содержание GC-оснований (не менее 50%). Для определения температуры отжига праймеров использовали on-line приложение OligoCalc URL (http://www.basic.northwestern.edu/biotools/oligocalc.html). Анализ возможности образования олигонуклеотидами вторичных структур, понижающих эффективность ПЦР, проверяли также с помощью программы VectorNTI v.8 (InforMax, США).
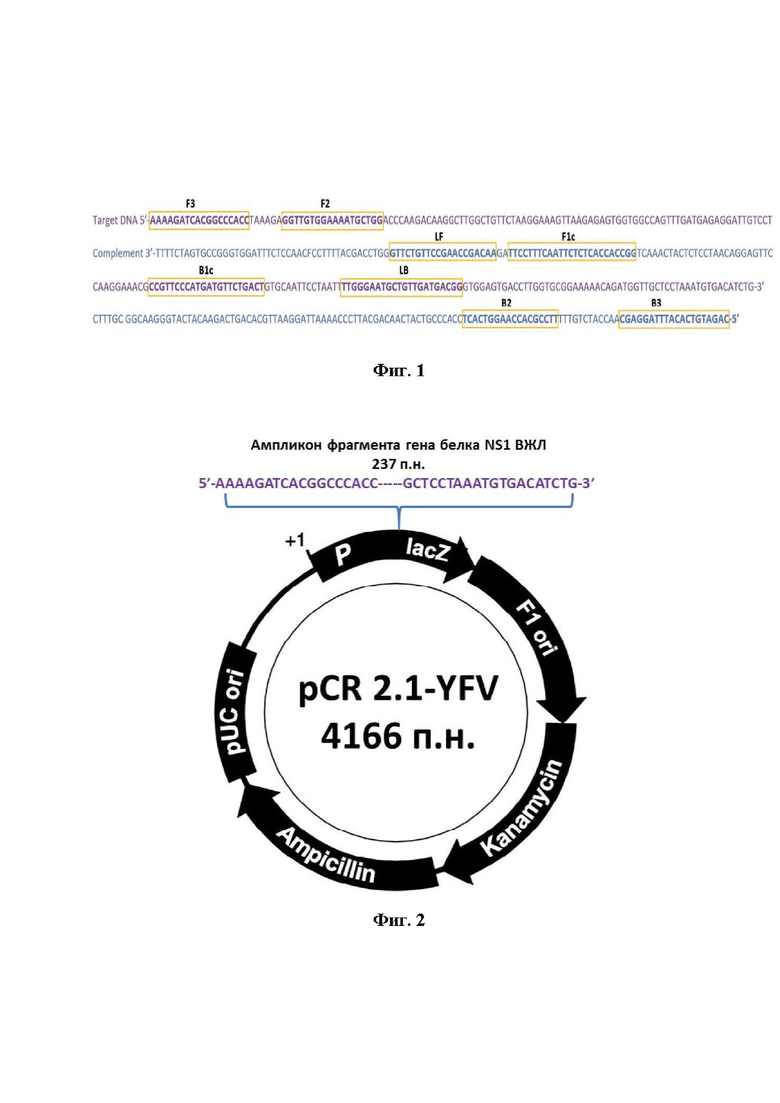
В результате были получены 6 олигонуклеотидных праймеров (производства ФБУН ГНЦ ВБ «Вектор» Роспотребнадзора), структура которых представлена в таблице 2. Области наложения праймеров на нуклеотидную последовательность гена белка NS1 ВЖЛ изображены на Фиг.1.
Таблица 2
Перечень разработанных олигонуклеотидных праймеров
Получение положительного контроля
Для получения ампликона для клонировали проводили ПЦР с использованием разработанных праймеров - F3 и B3, и кДНК ВЖЛ. Реакционная смесь для ПЦР из расчета на 1 реакцию:
5x Screen Mix-HS (Evrogen, Россия) - 5 мкл;
Праймер F3 (2 о.е.) - 1 мкл;
Праймер B3 (2 о.е.) - 1 мкл;
вода очищенная - 16 мкл;
Образец- 2 мкл.
Амплификацию проводили в термоциклере T100 («BioRad», США).
Температурно-временной режим амплификации
Для визуализации результатов ПЦР использовался метод гель-электрофореза в 2% агарозном геле. Для наблюдения за электрофорезом в пробу добавляют краситель, который передвигается со скоростью несколько большей, чем скорость движения наиболее подвижных макромолекул. Также добавляли ДНК-маркер для сопоставления размеров ампликонов. Гель-электрофорез проводили в течении 30 минут при постоянном напряжении 5 В/см в буфере 1х TAE, затем визуализировали результаты в трансиллюминаторе. Полученные ампликоны вырезались скальпелем и элюировались с помощью набора для элюции ДНК из агарозного геля BioSilica (Новосибирск, Россия), согласно инструкции производителя.
Для реакции клонирования использовали 2 мкл «свежнаработанного» ПЦР продукта и 1 мкл вектора pCR2.1-TOPO («Thermo Fisher Scientific», США) по инструкции производителя.
Подготовку компетентных клеток и трансформацию проводили по следующему протоколу:
1. Засеивали культуру E. coli штамм TOP10 («Thermo Fisher Scientific», США) в колбу с 10 мл среды LB (1% триптон, 1% хлорид натрия, 0,5% дрожжевой экстракт), далее ставили колбу в качалку при 37° на 180 об/мин на 2,5-3 часа до плотности 4-7+107 клеток/мл;
2. Охлаждали колбу во льду в течение 5 минут, затем разливали по 3 мл на пробу в стерильные пробирки, далее центрифугировали при 3000 об/мин 5 минут;
3. Среду сливали и к осадку добавляли 1 мл буфера I для трансформации (10mM MOPS (3-Морфолинопропансульфоновая кислота) (pH 7,0), 10mM рубидий хлористый, 5M гидроксид натрия), осторожно перемешивали и центрифугировали (3000 об/мин, 5 мин);
4. Жидкость сливали и к осадку добавляли 1 мл охлажденного буфера II для трансформации (0,1mM MOPS (pH 6,5), 0,05М кальций хлористый двуводный, 10M рубидий хлористый, 5M NaOH), далее пробирки помещали в лед на 15 мин, после этого центрифугировали (3000 об/мин, 5 мин);
5. Жидкость выливали и к осадку добавляли 200 мкл охлажденного буфера II для трансформации, 3 мкл ДМСО и пробу плазмидной ДНК, затем эту смесь помещали в лед на 30 минут;
6. После этого пробирки прогревали на водяной бане при 42°С на 30 с и затем охлаждали во льду 5 мин;
7. Затем добавляли 1 мл среды SOC (2% триптон, 0,5% дрожжевой экстракт, 10 мМ хлорид натрия, 2,5 мМ KCl, 10 мМ хлорид магния, 10 mM сульфат магния, 2 мМ глюкоза) и культивировали при 37° на качалке с перемешиванием 180 об/мин на час;
8. Далее центрифугировали (3000 об/мин, 5 мин), сливали среду, осадок ресуспендировали в 100 мкл среды SOC и засевали на чашки Петри с LB агаром (1%триптон, 1% хлорид натрия, 0,5% дрожжевой экстракт, 1,5% агар), содержащим ампициллин (100 мкг/мл).
Выделение плазмидной ДНК проводили с помощью набор diaGene для выделения плазмидной ДНК из бактерий («ДИАЭМ», Россия).
Для подтверждения правильной встройки целевого фрагмента проводили определение нуклеотидных последовательности. Секвенирование продуктов амплификации проводили по обеим цепям модифицированным методом Сэнгера на основе капиллярного электрофореза с помощью автоматического секвенатора 3130xl Genetic Analyzer («Applied Biosystems», США). Анализ полученных нуклеотидных последовательностей проводили с использованием программы UniproUGENE v. 1.30 и MEGA7.
Схема полученной рекомбинантной плазмиды pCR 2.1-YFV представлена на Фиг. 2.
Постановка реакции изотермической амплификации
Метод основывается на обратной транскрипции и петлевой изотермической амплификацией с использованием 6 праймеров, которые направлены на распознание 8 регионов таргетной кДНК. Bst ДНК-полимераза, обладающая способностью к расплетанию цепей, инициирует синтез, и два праймера (FIP и BIP) формируют петлеобразную структуру для облегчения последующих раундов амплификации. Для детекции продуктов амплификации применяется интеркалирующий флуоресцентный краситель и накопление флуоресцентного сигнала соответствует наличию в реакционной смеси специфических продуктов амплификации.
Для изотермической ПЦР использовали реакционную смесь следующего состава:
БиоМастер LAMP (2x) (Биолабмикс, Россия) - 12,5 мкл;
RNAscribe RT (Биолабмикс, Россия) - 1 мкл;
Праймер F3 (2 о.е.) - 1 мкл;
Праймер B3 (2 о.е.) - 1 мкл;
Праймер FIP (8 о.е.) - 1 мкл;
Праймер BIP (8 о.е.) - 1 мкл;
Праймер LF (2 о.е.) - 1 мкл;
Праймер LR (2 о.е.) - 1 мкл;
Интеркалирующий краситель EvaGreen (20х) (Biotium, США) - 1 мкл
Образец - 5 мкл.
Амплификацию и регистрацию результатов проводили на приборе CFX 96 (BioRad, США) по каналу FAM.
Программа изотермической амплификации
флуоресценции
Результаты интерпретируются на основании наличия (или отсутствия) пересечения кривой флуоресценции с установленной на соответствующем уровне пороговой линией (что соответствует наличию (или отсутствию) значения порогового цикла "Ct" в соответствующей графе в таблице результатов).
Результат подлежит учету в случае:
наличие положительного сигнала на канале FAM в пробах с положительным контрольным образцом (ПКО);
отсутствия положительного сигнала на канале FAM в пробах с отрицательным контрольным образцом (ОКО).
Результат считать положительным в случае, если кривая накопления флуоресценции для соответствующего образца имеет характерную «сигмовидную» форму и пересекает пороговую линию.
Статистическая обработка
Статистическую обработку данных осуществляли с использованием программного обеспечения Microsoft Excel.
Коэффициент вариации для повторяемости рассчитывали по формуле: CVp, % = Ct (станд.отклон) / Ct (ср.знач.) × 100% для 5 проб.
Коэффициент вариации для воспроизводимости рассчитывали по формуле: CVp, % = Ct (станд.отклон) / Ct (ср.знач.) ×100% для 10 проб.
Изобретение иллюстрируется следующими графическими материалами.
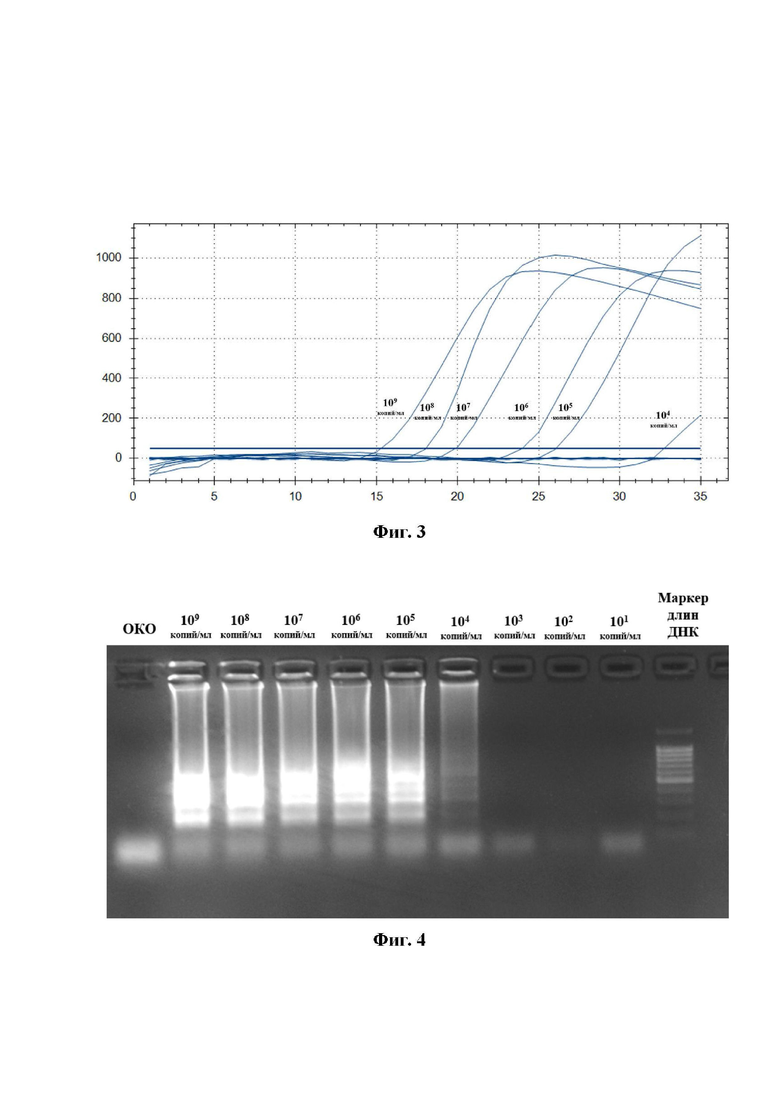
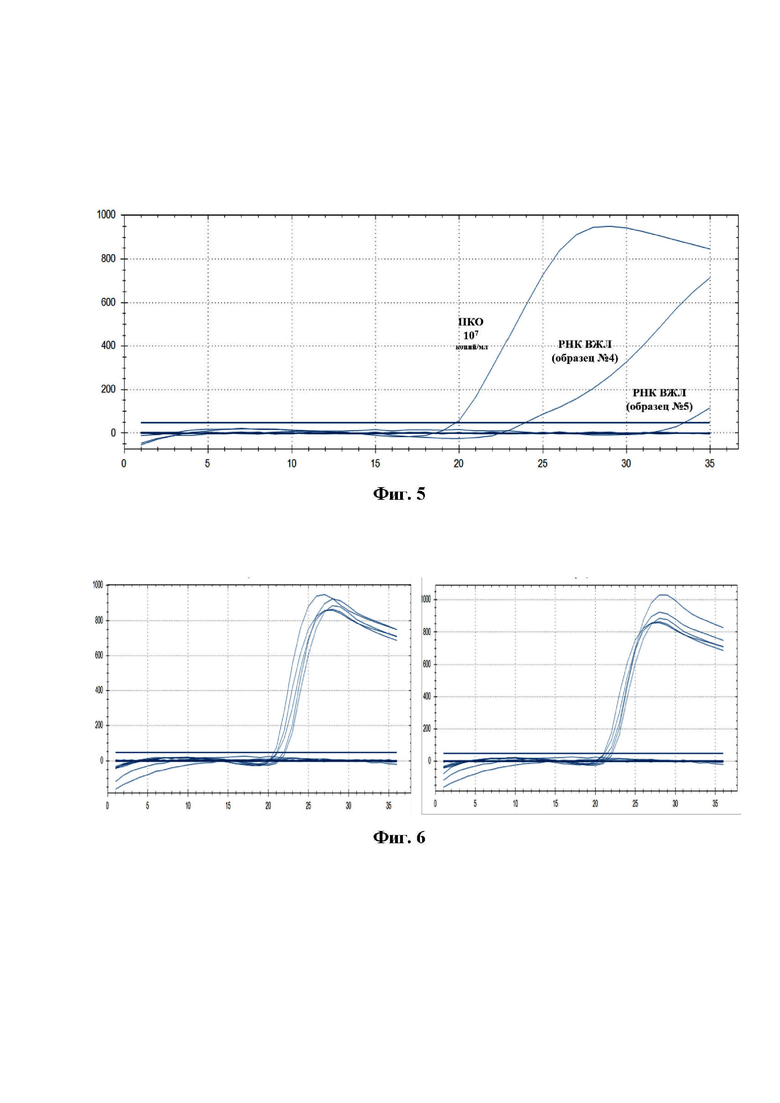
На Фиг. 1. олигонуклеотидная последовательность клонированного в плазмиду ампликона ВЖЛ с указанием областей локализации праймеров на фрагменте гена белка NS1 ВЖЛ штамма Asibi (GenBank: MF405338.1). На Фиг. 2. упрощенная схема плазмидной ДНК pCR 2.1-YFV с указанием места вирусоспецифической вставки для контроля аналитической чувствительности разработанного набора праймеров. На Фиг. 3. представлен результат изотермической амплификации с детекцией в режиме реального времени (на приборе CFX96) для оценки чувствительности разработанного набора олигонуклеотидных праймеров. На Фиг. 4. изображен результат изотермической амплификации с электрофоретической детекцией в 2% агарозном геле для подтверждения результатов оценки чувствительности с детекцией в режиме реального времени. На Фиг. 5. представлен результат оценки специфичности к генетическому материалу ВЖЛ разработанного набора праймеров. На Фиг. 6. показана оценка повторяемости и воспроизводимости результатов изотермической амплификации при использовании набора праймеров. На Фиг. 7 представлены последовательности олигонуклеотидных праймеров специфичных к кДНК ВЖЛ SEQ ID: NO: 1-6: праймеры внешние - F3 и B3, внутренние FIP и BIP, петлевые праймеры - LF и LB.
Пример 1. Проверка аналитической чувствительности разработанного набора олигонуклеотидных праймеров для выявления РНК ВЖЛ методом изотермической амплификации
Контроль аналитической чувствительности (Предела обнаружения (LoD) осуществляли проведением изотермической амплификации с обратной транскрипцией и флуоресцентной детекцией, где в качестве исследуемых образцов использовали последовательные разведения pCR 2.1-YFV с шагом 10 от 109 копий/мл до 10 копий/мл. В качестве отрицательного контрольного образца (ОКО) использовали воду очищенную. Результат оценки аналитической чувствительности представлен в таблице 5, на Фиг. 3 и Фиг. 4.
Таблица 5
Результат оценки чувствительности изотермической амплификации с использованием разработанных олигонуклеотидных праймеров
Образцы, содержащие pCR 2.1-YFV в концентрациях от 109 копий/мл до 104 копий/мл определены как положительные. Образцы содержащие pCR 2.1-YFV в концентрациях от 103 копий/мл до 10 копий/мл и ОКО определены как отрицательные. Установлена аналитическая чувствительность (предел детекции) - 104 копий/мл.
Пример 2. Проверка специфичности разработанного набора олигонуклеотидных праймеров для выявления РНК ВЖЛ методом изотермической амплификации
Для определения диагностической специфичности использовали образцы сыворотки крови искусственно контаминированные РНК ВЖЛ, вирусов денге (1-4), вируса клещевого энцефалита и вируса Зика. В качестве положительного контрольного образца (ПКО) использовали pCR 2.1-YFV (107 копий/мл), в качестве отрицательного ОКО и ДНК человека, выделенная из крови здоровых доноров.
Подготовка анализируемых проб. В соответствии с МУ 1.3.2569-09 «Организация работы лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности» для проведения процедуры обеззараживания 100 мкл материала помещали в пробирку объемом 1,5 мл, добавляли 500 мкл лизирующего буфера, на основе 6 М гуанидинизотиоционата и фенола (1:1) и инкубировали 20 мин при температуре 65 °С.
Таблица 6
Результат оценки специфичности изотермической амплификации с использованием разработанных олигонуклеотидных праймеров
(107 копий/мл)
После проведения процедуры обеззараживания производили выделение РНК в соответствии с Инструкцией по применению комплекта реагентов для выделения («РИБО-преп» РУ № ФСР 2008/03147, производства ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия). В качестве пробы использовали 100 мкл материала после проведения процедуры обеззараживания.
Результат оценки специфичности изотермической амплификации с использованием разработанных праймеров представлен в табл. 6 и Фиг. 5.
Пробы, содержащие генетический материал ВЖЛ определены как положительные (совпадение 100%). Пробы, не содержащие генетический материал ВЖЛ определены как отрицательные (совпадение 100%).
Пример 3. Оценка повторяемости и воспроизводимости результатов изотермической амплификации с использованием разработанного набора праймеров
Для оценки повторяемости 5 образцов pCR 2.1-YFV с концентрацией 107 копий/мл одновременно протестированы одним оператором (Фиг. 6).
Таблица 7
Оценка повторяемости и воспроизводимости изотермической амплификации с использованием разработанных праймеров
обр.
FAM
Ct FAM
Оператор 1
Оператор 2
Коэффициент вариации для повторяемости рассчитывали по формуле: CVp, % = Ct (станд.отклон) / Ct (ср.знач.) × 100 % для 5 проб.
Для оценки воспроизводимости во второй день на другом приборе дополнительно протестированы 5 образцов pCR 2.1-YFV с концентрацией 107 копий/мл другим оператором (Фиг. 6).
Расчетные коэффициенты вариации для повторяемости и воспроизводимости
Коэффициент вариации для воспроизводимости рассчитывали по формуле: CVp, % = Ct (станд.отклон) / Ct (ср.знач.) ×100 % для 10 проб.
Результаты расчетов представлены в таблице 7 и таблице 8.
Максимальный коэффициент вариации для повторяемости составил 2,44%, коэффициент вариации для воспроизводимости 2,23%.
Источники научно-технической и патентной информации
1. Zeller HG, Schuffenecker I. West Nile virus: an overview of its spread in Europe and the Mediterranean basin in contrast to its spread in the Americas. Eur J Clin Microbiol Infect Dis. 2004 Mar; 23(3):147-56. doi: 10.1007/s10096-003-1085-1.
2. Florian Douam, Alexander Ploss. Yellow Fever Virus: Knowledge Gaps Impeding the Fight Against an Old Foe // J. Emerging Microbes & Infections. 2018. Vol. 28. P. 913-928 doi: 10.1016/j.tim.2018.05.012.
3. Ganushkina L.A. et al. Detection of Aedes aegypti, Aedes albopictus, and Aedes koreicus in the Area of Sochi, Russia // Vector Borne Zoo notic Dis. 2016. Vol. 16, N 1. P. 58-60. doi: https://doi.org/10.1089/vbz.2014.1761.
4. Domingo C, Charrel RN, Schmidt-Chanasit J, Zeller H, Reusken C. Yellow fever in the diagnostics laboratory. Emerg Microbes Infect. 2018 Jul 12;7(1):129. doi: 10.1038/s41426-018-0128-8.
5. WHO [Электронный ресурс]. URL: https://www.who.int/emergencies/disease-outbreak-news/item/2022-DON361 (Дата обращения 14.03.2024)
6. WHO [Электронный ресурс]. URL: https://www.who.int/emergencies/disease-outbreak-news/item/yellow-fever-west-and-central-africa (Дата обращения 14.03.2024)
7. WHO [Электронный ресурс]. URL: https://www.who.int/emergencies/disease-outbreak-news/item/2022-DON367 (Дата обращения 14.03.2024)
8. WHO. [Электронный ресурс]. URL: https://www.who.int/csr/don/archive/disease/yellow_fever/ru/ (дата обращения 14.03.2024).
9. Drosten C., Kummerer B.M., Schmitz H., Gunther S. 2003. Molecular diagnostics of viral hemorrhagic fevers. Antiviral Res 57:61- 87. https://doi.org/10.1016/S0166 3542(02)00201-2
10. Кривошеина Е.И., М.Ю. Карташов, Е.В. Найденова Современные лабораторные методы выявления возбудителя желтой лихорадки. Проблемы особо опасных инфекций. 2021; 2:24-32. DOI: 10.21055/0370-1069-202-2-21-32
11. Nunes, M.R. et al. Analysis of a Reverse Transcription Loop-mediated Isothermal Amplification (RT-LAMP) for yellow fever diagnostic. J. Virol. Methods226, 40-51 (2015).
12. Kwallah, A. et al. A real-time reverse transcription loop-mediated isothermal amplification assay for the rapid detection of yellow fever virus. J. Virol. Methods 193, 23-27 (2013).
13. Патент США №11473155, МПК C12Q1/6806, опубл. 18.10.2022 г. (прототип).
ПРИЛОЖЕНИЕ
Перечень последовательностей, представленных
по стандарту ST26 ВОИС
--->
<ST26SequenceListing dtdVersion="V1_3" fileName="Набор олигонуклеотидных праймеров
для выявления РНК вируса желтой лихорадки (ВЖЛ) методом
изотермической амплификации в режиме реального времени.xml" softwareName=
"WIPO Sequence" softwareVersion="2.3.0"
productionDate="2024-03-16">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>1</ApplicationNumberText>
<FilingDate>2024-03-16</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>1</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>1</ApplicationNumberText>
<FilingDate>2024-03-16</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">Федеральное бюджетное учреждение
науки «Государственный научный центр вирусологии и
биотехнологии «Вектор» Федеральной службы по надзору в сфере
защиты прав потребителей и благополучия человека
(ФБУН ГНЦ ВБ «Вектор» Роспотребнадзора)</ApplicantName>
<ApplicantNameLatin>State Research Center of Virology and Biotechnology "Vector"
</ApplicantNameLatin>
<InventionTitle languageCode="ru">Набор олигонуклеотидных праймеров
для выявления РНК вируса желтой лихорадки (ВЖЛ) методом
изотермической амплификации в режиме реального времени</InventionTitle>
<SequenceTotalQuantity>6</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q13">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aaaagatyacggcycacc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cagatgtcacatttaggagc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>47</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..47</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q15">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggccaccactctyttaactttccttttttggttgtggaaaatgctgg<
/INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>43</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..43</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q16">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccgttcccatgatgttctgacttttttccgcaycaaggtcact</
INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q17">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>arcagccaagccttgtcttg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q18">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttgggaatgctgttgatgacgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---


Изобретение относится к биотехнологии и вирусологии. Предложен набор олигонуклеотидных праймеров для выявления РНК вируса желтой лихорадки в клинических образцах человека (сыворотка крови) и вируссодержащих культуральных жидкостях для идентификации завозных случаев желтой лихорадки на территории Российской Федерации, который содержит 6 праймеров: внешние - F3 и B3, внутренние FIP и BIP, петлевые праймеры - LF и LB, имеющие нуклеотидные последовательности SEQ ID NO: 1-6, соответственно. Набор олигонуклеотидных праймеров обеспечивает петлевую изотермическую амплификацию генетического материала ВЖЛ. 7 ил., 8 табл., 3 пр.
Набор олигонуклеотидных праймеров для выявления РНК вируса желтой лихорадки (ВЖЛ) методом изотермической амплификации в режиме реального времени, содержащий 6 праймеров: внешние - F3 и B3, внутренние FIP и BIP, петлевые праймеры - LF и LB, имеющие следующие нуклеотидные последовательности SEQ ID NO: 1-6, соответственно.
| CN 102776297 A, 14.11.2012 | |||
| CN 102191338 A, 21.09.2011 | |||
| Paban Kumar Dash et al., Development of a SYBR green I based RT-PCR assay for yellow fever virus: application in assessment of YFV infection in Aedes aegypti, Virology Journal volume 9, Article number: 27 (2012), весь документ | |||
| НАБОР ОЛИГОНУКЛЕОТИДНЫХ ПРАЙМЕРОВ И ФЛУОРЕСЦЕНТНО-МЕЧЕНОГО ЗОНДА ДЛЯ ИДЕНТИФИКАЦИИ РНК ВИРУСА ЛИХОРАДКИ ДОЛИНЫ РИФТ МЕТОДОМ ОТ ПЦР В РЕАЛЬНОМ ВРЕМЕНИ | 2014 |
|
RU2552795C1 |
| ИНСТРУКЦИЯ ПО ПРИМЕНЕНИЮ набора | |||
Авторы
Даты
2024-08-06—Публикация
2024-04-27—Подача