Изобретение относится к области молекулярной диагностики, в частности, к выявлению ДНК вируса африканской чумы свиней (АЧС) с помощью метода амплификации нуклеиновых кислот, а именно петлевой изотермической амплификации (LAMP).
Изобретение может быть использовано в ветеринарной лабораторной диагностике для выявления ДНК вируса АЧС в биологическом материале от животных - цельной/плазме/сыворотке крови, мазках со слизистых оболочек, суспензиях тканей (лимфоузлы, миндалины, селезенка, легкие, печень).
Африканская чума свиней - высококонтагиозная вирусная болезнь свиней, характеризующаяся лихорадкой, цианозом кожи и обширными геморрагиями во внутренних органах. Относится к особо опасным заразным болезням животных, согласно списку МЭБ по Международной классификации заразных болезней животных, которые определяются как "заразные (трансмиссивные) болезни, которые имеют способность к опасному и быстрому распространению безотносительно к государственным границам, сопровождаются серьезными последствиями в области общественной экономики и здравоохранения, имеют важное значение в международной торговле животными и продуктами животноводства".
Возбудитель африканской чумы свиней - крупный ДНК-содержащий вирус семейства Asfarviridae (диаметр вириона около 200 нм). Вирус имеет округлую или икосаэдрическую форму, структура вириона сложная, выявляются несколько слоев - внешняя суперкапсидная оболочка, происходящая из клеточной и приобретаемая в процессе выхода из клетки почкованием, икосаэдральный капсид, состоящий из субъединиц-капсомеров и сердцевина из слоя фибриллярных компонентов и нуклеотида. В виду особенностей строения вирус АЧС очень устойчив к факторам различной природы, в связи с чем его выживаемость в объектах среды и продуктах свиного происхождения необычно высока [1, 2].
Вирус обладает высокой степенью патогенности (смертность среди животных достигает 100%). Заболевание является эндемичным для многих стран Южной Африки. В 2007 г. вирус АЧС был занесен в Кавказский регион, изначально в Грузию, затем в Армению, Азербайджан и во многие регионы Российской Федерации и Белоруссию. С начала 2014 г. вспышки африканской чумы свиней фиксируются во многих Европейских странах (Латвия, Литва, Эстония, Польша, Венгрия, Чехия, Румыния, Болгария, Словакия, Сербия, Бельгия и др.). В 2018 году, когда АЧС получила распространение в Китае, стране с огромным поголовьем свиней, ситуация значительно ухудшилась, позднее АЧС распространилась и в других странах Азиатского континента, в том числе Монголии, Вьетнаме, Камбодже, Северной Корее, Мьянме, Лаосе, Филиппинах [3].
Разработанные методы лечения и специфической профилактики болезни малоэффективны, поэтому распространение АЧС наносит огромные экономические потери, обусловленные проведением ветеринарно-санитарных и карантинных мероприятий и убоя всего естественно восприимчивого поголовья на территории очага инфекции.
Несмотря на многолетней опыт и имеющиеся подробные рекомендации по лабораторной диагностике, идентификация вируса АЧС традиционными методами (по его биологическим свойствам) требует значительных затрат и времени. Для лабораторной диагностики Всемирной организацией здравоохранения животных (МЭБ) рекомендуются молекулярные методы, как обладающие высокой чувствительностью и специфичностью, в том числе ПЦР (полимеразная цепная реакция) с детекцией продуктов амплификации в агарозном геле или ПЦР в режиме реального времени с гибридизационно-флуоресцентными зондами. Использование подобных ПЦР методик в диагностике АЧС значительно повысило эффективность своевременной расшифровки данной этиологии [4, 5]. ПЦР имеет хорошие аналитические и диагностические характеристики, однако требует дорогостоящего оборудования, а исследование занимает достаточно много времени (не менее 4-6 часов, включая этап экстракции нуклеиновых кислот). Новые методы изотермической амплификации появились в начале 21 века и успешно применяются для выявления широкого круга возбудителей инфекционных болезней. Так метод петлевой амплификации, LAMP, предложенный японским ученым Tsugunori Notomi в 2000 году [6], основывался на использовании праймеров к шести различным участкам целевой области генома. Впоследствии, метод был усовершенствован путем добавления пары петлевых праймеров [7], обеспечивающих дополнительную амплификацию с петель образующихся продуктов LAMP, что повысило эффективность реакции. Преимуществом изотермических методов является сокращение времени амплификации от нескольких часов до 15-30 минут. Другим важным преимуществом изотермических методов является возможность их применения в условиях ограниченных ресурсов без использования сложного оборудования. Изотермическая реакция может идти при постоянной температуре в термостате, а детекция результатов возможна визуальным способом, например, за счет помутнения раствора из-за появления осадка пирофосфата магния [8] или изменения цвета pH-чувствительных красителей [9].
Для выявления вируса АЧС предложено несколько вариантов методик изотермической амплификации, основанных на различных технологиях (LAMP, RPA, RAA-CAS12a) однако в большинстве случаев детекция результатов амплификации осуществляется с использованием неспецифической без-зондовой детекции (например с использованием интеркалирующих и рН зависимых красителей [10, 11] либо иммунохроматографических полосок [12]). Во многих случаях заявленная аналитическая чувствительность подобных быстрых методик достаточно низкая, так чувствительность RPA с детекцией на иммунохроматографических полосках составляет 150 копий на реакцию [13], хотя в отдельных случаях авторам удается добиться чувствительности сравнимой с чувствительностью метода ПЦР, так для LAMP c колориметрической детекцией авторами [14] заявлена чувствительность до 10 копий на реакцию. Колориметрические методы имеют преимущества по сравнению с другими методами неспецифической детекции, поскольку они более чувствительны, а также более устойчивы к наличию непрозрачных веществ в реакционной смеси, в отличие от турбидиметрического анализа.
Однако общим основным недостатком использования всех неспецифических методов обнаружения является повышенная вероятность неспецифических реакций [14] Флуоресцентные зонды, такие как молекулярные маяки или TaqMan зонды [15, 16], широко используются для детекции накопления продуктов амплификации при ПЦР диагностике. Данные олигонуклеотидные зонды имеют флуорофор и гаситель флуоресценции, флуоресценция испускается при отдалении молекулы-гасителя от флуорофора. В случае молекулярного маяка это происходит при разрушении шпилевидной структуры зонда при его гибридизации с последовательностью-мишенью, в случае с TaqMan-зондом, зонд разрушается при амплификации за счет экзонуклеазной активности Taq ДНК полимеразы и гаситель отщепляется от флуорофора. Однако оба типа зондов не могут использоваться для детекции результатов LAMP. Молекулярные маяки самопроизвольно разворачиваются при температуре реакции LAMP (от 60 до 65°C), что повышает фоновый сигнал, а TaqMan зонды не могут быть разрушены Bst ДНК полимеразой не имеющей экзонуклеазной активности, в отличие от Taq ДНК полимеразы.
Наиболее близким аналогом заявленного изобретения по сути и назначению является метод петлевой изотермической амплификации с использованием интеркалирующих красителей, разработанный авторами патента RU 2710065 C1. Данный метод является экспресс тестом, позволяющим провести амплификацию в течение 30-40 минут, однако его применение в условиях хорошо оснащенных лабораторий не оправдано, из-за невозможности контролировать потери нуклеинового материала и очистку от ингибирующих реакцию амплификации веществ при экстракции ДНК. Более того амплификация в присутствии интеркалирующих красителей может давать ложноположительные результаты из-за неспецифичных реакций, что ухудшает диагностические характеристики метода. Использование внутреннего контрольного образца с этапа экстракции ДНК, предусмотренное в нашем изобретении, позволяет контролировать потери нуклеинового материала и ингибирование реакции амплификации, что значительно улучшает диагностическую чувствительность метода и снижает количество ложно-отрицательных результатов.
С целью устранения вышеуказанных недостатков задачей настоящего изобретения является разработка высокочувствительного и экспрессного способа выявления присутствия ДНК вируса АЧС при помощи петлевой изотермической амплификации с гибридизационно-флуоресцентными зондами. Разработанный способ является быстрой альтернативой ПЦР и позволяет мультиплексировать реакции выявления ДНК вируса АЧС и ДНК внутреннего контрольного образца в одной пробирке, используя различные флуоресцирующие метки. В качестве зондов использовали модифицированные внутренние праймеры FIP, содержащие на 5‘ конце гаситель флуоресценции в дуплексе с небольшим олигонуклеотидом, содержащим на 3’ конце флуорофор. Также в отличие от способа предложенного в патенте RU 2 710 065 C1, использующего в качестве мишени фрагмент гена кодирующего белок р22 вируса АЧС, в данной работе олигонуклеотидные праймеры подобраны к хорошо изученной и часто используемой для разработки ПЦР методик выявления вируса АЧС мишени - фрагменту гена мажорного капсидного белка p72 [17, 18, 19, 20].
Задача по разработке высокочувствительного, специфичного и быстрого метода выявления вируса АЧС была решена благодаря созданию олигонуклеотидных праймеров комплементарных участку гена p72 вируса АЧС, а также созданию праймеров комплементарых участку правого плеча генома бактериофага лямбда и подбору условий для проведения их совместной петлевой изотермической амплификации, обеспечивающей получение результата в течение 20-30 минут.
Сущность изобретения заключается в новом подходе обнаружения ДНК вируса АЧС с помощью проведения модифицированной петлевой изотермической амплификации с использованием разработанных олигонуклеотидных праймеров ( ASF-F3, ASF-В3, ASF-F, ASF-BL, ASF-FIP, ASF-BIP), фланкирующих специфический фрагмент генома вируса АЧС, олигонуклеотидных праймеров (IC-F3, IC-В3, IC-F, IC-BL, IC-FIP, IC-BIP) фланкирующий специфический фрагмент бактериофага лямбда, а также зондов ASF-FIP- BHQ1/ ASF-R6G, IC-FIP- BHQ1/ IC- FAM на основе внутренних праймеров FIP. Использованный в данном изобретении подход основан на применении FIP модифицированных праймеров, несущих на 5’ конце гаситель флуоресценции, в сочетании с олигонуклеотидами, комплементарными 5’ участкам FIP праймеров и несущим на 3’ конце флуорофор, как описано ранее авторами [24].
Представленный способ включает последовательности олигонуклеотидных праймеров, имеющих следующую нуклеотидный состав:
ASF-F3 ATTACGTCTTATGTCCAGA,
ASF-В3 AGACTGGATATAAGCACTTG,
ASF-F TCACAATATCCAAACAGCAGGT,
ASF-BL TAAGAATAGGTTTGCTYTGGTG,
ASF-FIP ACCAACCCGAAATTCCTTTCACTTTTTGCGTCCGTAATAGRGTRATA,
ASF-BIP GTTCGCTCGTATCATTTTCATTTTTAGGTATCGGTGGAGGGAACY,
IC-F3 AAACGCAACGAGGCTCTA,
IC-В3 GGATTTGTTCAGAACGCTC,
IC-FL ACAGCTTCCGCTGTCTTCTCA,
IC-BL GTTGACGACGACATGGCTC,
IC-FIP CCTGCTGATCTGCGACTTATTTCGAGAGTGCGTTGCTTAAC,
IC -BIP GGATTCCAAAGTTCTCAATGCTTTTGAGAATCGCAGCAACTTGT
ASF-FIP- BHQ1/ ASF-R6G
BHQ1-ACCAACCCGAAATTCCTTTCACTTTTTGCGTCCGTAATAGRGTRATA / GTGAAAGGAATTTCGGGTTGGT-R6G,
IC-FIP- BHQ1/ IC- FAM
BHQ1-CCTGCTGATCTGCGACTTATTTCGAGAGTGCGTTGCTTAAC /
ATAAGTCGCAGATCAGCAGG-FAM.
Способ выявления ДНК вируса АЧС основан на экстракции ДНК из биологического материала совместно с ДНК экзогенного контрольного образца (бактериофага лямбда) и проведении амплификации полученной ДНК в режиме «реального времени» изотермическим способом. Амплификация фрагмента ДНК бактериофага лямбда при проведении исследования будет свидетельствовать об отсутствии ингибирования реакции и потерь ДНК при экстракции, что позволит исключить ложноотрицательные результаты.
С полученными на этапе экстракции пробами ДНК проводится изотермическая петлевая амплификация ДНК при помощи Bst ДНК полимеразы и набора специфичных праймеров олигонуклеотидов (ASF-F3, ASF-В3, ASF-F, ASF-BL, ASF-FIP, ASF-BIP, IC-F3, IC-В3, IC-F, IC-BL, IC-FIP, IC-BIP), включая меченые гасителями праймеры, связанные с флуоресцентно-меченными олигонуклеотидами (ASF-FIP-BHQ1/ ASF-R6G, IC-FIP- BHQ1/ IC- FAM). При амплификации происходит вытеснение флуоресцентно-меченого олигонуклеотида от праймера-гасителя, что приводит к нарастанию интенсивности флуоресценции.
Детекция флуоресцентного сигнала осуществляется с помощью амплификатора с системой детекции флуоресцентного сигнала. Таким образом при исследовании одновременно в одной пробирке проводятся две реакции - амплификация ДНК вируса АЧС и амплификация ДНК бактериофага лямбда, результаты регистрируются по двум каналам флуоресцентной детекции.
Для разработки праймеров использовали нуклеотидные последовательности фрагментов геномов изолятов вируса АЧС из баз данных NCBI, включая отечественные опубликованные последовательности. Последовательности выравнивали относительно друг друга в программах Mega-X (алгоритм ClustalW), Ugene (алгоритм MUSCLE) для выявления специфичных и консервативных участков. Затем на выбранных достаточно консервативных для вируса АЧС участках генома выбирали праймеры для LAMP. Праймеры проверяли на отсутствие значимой гомологии с нуклеотидными последовательностями всех нецелевых организмов из базы данных NCBI, которая включает в себя нуклеотидные последовательности GenBank + EMBL + DDBJ + PDB, использовали ресурс Basic Local Alignment Search Tool BLAST [22]. Также оценивали наличие полиморфизмов в областях праймеров для различных последовательностей целевого вируса, при необходимости вносили в олигонуклеотидные праймеры вырождения, чтобы учесть значимые варианты. Менее значимые полиморфизмы, расположенные внутри праймеров или на 5`-конце для праймеров F3, B3 и фрагментов F2, B2 праймеров FIP, BIP или на 3`-конце фрагментов F1c, B1c праймеров FIP, BIP оставляли без вырождения. Праймеры для LAMP подбирали с учетом следующих критериев: специфичность; температура плавления праймеров; состав праймеров; расстояние между праймерами; отсутствие значимых шпилек; отсутствие значимых димеров на 3`- концах праймеров; отсутствие значимых димеров с другими праймерами; длина праймера; GC-состав; в составе праймеров избегали динуклеотидных повторов внутри праймеров, а также повторов одинаковых нуклеотидов больше 3 раз подряд.
Специфичность проверяли с использованием базы данных нуклеотидных последовательностей NCBI [22]. Температуру плавления праймеров оценивали с помощью онлайн-программы Oligo Calc: Oligonucleotide Properties Calculator [23]. Для внутренних участков праймеров (F1c, B1c, FL, BL) она должна быть на несколько градусов выше, чем для внешних (F3, B3, F2, B2). Отсутствие значимых вторичных структур (шпилек и димеров) проверяли с использованием программы PrimerSelect (Dnastar, Inc., Madison, WI). Для детекции результатов LAMP использовали модифицированные праймеры, образующие дуплекс с олигонуклеотидами несущими флуоресцентную метку. В образуемом перед реакцией амплификации дуплексе модифицированного праймера и флуоресцентно меченного олигонуклеотида флуорофор и гаситель находятся в непосредственной близости. В процессе петлевой амплификации Bst ДНК полимераза вытесняет флуоресцентно меченный олигонуклеотид с 5’ конца модифицированного праймера, что приводит к накоплению флуоресцентного сигнала в реакционной смеси.
Использование дуплексов меченного гасителем праймера с комплементарным олигонуклеотидом меченным флуорофором, позволяет следить за реакцией изотермической амплификации за счет накопления флуоресценции при включении модифицированного праймера в процесс амплификации с последующим вытеснением комплементарного олигонуклеотида Bst ДНК полимеразой.
Основная характеристика рассчитанных олигонуклеотидов представлена в таблице 1.
* Обозначения вырожденных нуклеотидов: Y = C или T, R =A или G.
Готовят 2,5 x смесь LAMP, содержащую следующие компоненты: праймеры для амплификации ДНК вируса АЧС и ДНК бактериофага лямбда (табл. 1) в следующих концентрациях ASF-FIP, IC-FIP - по 1.0 μM, ASF-BIP, IC-BIP - по 2.5 μM, ASF-F3, ASF-B3, IC-F3, IC-B3 - по 0.5 μM, ASF-FL, IC-FL, ASF-BL, IC-BL - 1.0 μM, зондов-праймеров ASF-FIP-BHQ1, IC-FIP-BHQ1 по 1.5 μM, ASF-R6G , IC -FAM - 2.0 μM, 1,4 мМ каждого дНТФ (Синтол, Россия), стерильная деионизованная вода.
Для проведения LAMP предварительно осуществляют экстракцию ДНК из исследуемых проб в присутствии добавленного перед экстракцией бактериофага лямбда (из коллекции НИЦ "Курчатовский институт" ГосНИИгенетика) до концентрации 10^5 - 10^6 копий/мл. Для экстракции ДНК могут использоваться комплекты реагентов «НК-сет-ОД», «НК-сорб-ОД» ОД-Тест или другие комплекты реагентов, соответствующие по назначению к исследуемому биологическому материалу.
Параллельно с ДНК пробами, экстрагированными из биологического материала от животных, исследуют контрольные образцы - «К+», содержащий ДНК вируса АЧС, «К-» не содержащий ДНК вируса АЧС и бактериофага лямбда, а также контрольный образец этапа экстракции нуклеиновых кислот «ОК», содержащий ДНК бактериофага лямбда и не содержащий ДНК вируса АЧС.
Анализ LAMP предпочтительно проводят в объеме реакционной смеси 25 мкл, содержащей 10 мкл ДНК-пробы; 5 мкл 5х-буфера для LAMP (Гентерра, Россия); 8 ед.а. Bst ДНК полимеразы (Гентерра, Россия); 10 мкл 2,5 x LAMP смеси; конечный объем 25 мкл.
В отдельной пробирке готовят реакционную смесь на необходимое количество образцов, в число которых входит отрицательный контроль экстракции (ОК), отрицательный и положительный контроли амплификации (К-, К+) плюс запас на одну реакцию.
Компоненты реакционной смеси смешивают непосредственно перед проведением LAMP-исследования. Реакционную смесь аккуратно перемешивают и вносят в подготовленные для амплификации пробирки по 15 мкл. Затем в пробирки вносят по 10 мкл проб ДНК, полученных в результате экстракции из исследуемых образцов, ставят контрольные реакции - положительный контроль (К+) - в пробирку с реакционной смесью вносят 10 мкл образца К+; отрицательный контроль (К-) - в пробирку с реакционной смесью вносят 10 мкл образца К-; отрицательный контроль экстракции (ОК) - в пробирку с реакционной смесью вносят 10 мкл пробы, экстрагированной из ОК. Общий объем реакции составляет 25 мкл.
Запрограммировать амплификатор с системой флуоресцентной детекции сигнала (например, Rotor-Gene Q (QIAGEN GmbH), Rotor-Gene 6000 (Corbett Research), CFX 96 (Bio-Rad), ДТпрайм (ДНК Технология)) для выполнения программы амплификации и детекции флуоресцентного сигнала - 65°С, 30 сек, детекция после каждого цикла на каналах FAM/Green, HEX/Yellow, установить 60 циклов. Установить пробирки в ячейки реакционного модуля прибора. По завершении амплификации проводят анализ полученных данных с помощью программного обеспечения прибора, используемого для проведения амплификации. Анализируют кривые накопления флуоресцентного сигнала по каналам FAM/Green и HEX/Yellow. Результаты интерпретируются на основании наличия (или отсутствия) пересечения кривой флуоресценции с установленной на соответствующем уровне пороговой линией, что определяет наличие (или отсутствие) для данной пробы ДНК значения порогового цикла (Сt) в соответствующей графе таблицы результатов.
Принципы интерпретации результатов следующие:
Обнаружена ДНК вируса АЧС если определено значение Ct на канале для флуорофора HEX/Yellow, значение порогового цикла (Сt) по каналу для флуорофора FAM/Green может быть определено или отсутствовать. Не обнаружена ДНК вируса АЧС если отсутствует значение Ct на канале для флуорофора HEX/Yellow, при этом значение порогового цикла (Сt) по каналу для флуорофора FAM/Green определено менее граничного значения (Сt ≤45). Результат является невалидным если отсутствует значение Ct на канале для флуорофора HEX/Yellow, при этом значение порогового цикла (Сt) по каналу для флуорофора FAM/Green не определено или определено более граничного значения (Сt >45).
В случае получения невалидного или сомнительного результата необходимо провести повторное исследование образца, начиная с этапа экстракции ДНК. В случае повторения результата, рекомендуется повторное взятие материала для анализа.
Результаты подлежат учету только если получены корректные результаты для контролей этапов экстракции и амплификации ДНК : K+ - по каналу флуоресценции FAM/Green значение порогового цикла не учитывается, по каналу флуоресценции HEX/Yellow определено значение Ct <40; K - по обоим каналам флуоресценции FAM/Green, HEX/Yellow значения пороговых циклов (Сt) отсутствуют; ОК - по каналу флуоресценции FAM/Green значение порогового цикла определено Ct <40, по каналу флуоресценции HEX/Yellow значение Ct отсутствует.
Если для положительного контроля K+ значение порогового цикла (Сt) по каналу для флуорофора HEX/Yellow отсутствует или превышает граничное значение необходимо повторить исследование для всех образцов (начиная с этапа экстракции ДНК). Если для отрицательного контроля (К-) определено значение порогового цикла (Сt) по каналам для флуорофоров FAM/Green, HEX/Yellow или для отрицательного контроля экстракции (ОК) определено значение порогового цикла (Сt) по каналу для флуорофора HEX/Yellow - высока вероятность контаминации лаборатории или исследуемых образцов продуктами амплификации, необходимо предпринять меры по выявлению и ликвидации источника контаминации и повторить исследование для всех образцов.
Все значения пороговых циклов, Ct, учитываются только для сигналов, поднявшихся выше фонового уровня шума и имеющих вид экспоненциальных кривых. Если для исследуемого образца определено значение порогового цикла (Ct), при этом на графике флуоресценции отсутствует участок характерного экспоненциального подъема необходимо проверить правильность выбранного уровня пороговой линии или параметров расчета базовой линии. Если результат получен при правильных настройках пороговой и базовой линий, исследование для этого образца необходимо повторить.
Сущность заявленного изобретения пояснена примерами осуществления изобретения.
Пример 1. Определение специфичности петлевой изотермической амплификации с гибридизационно-флуоресцентными зондами для выявления генома вируса АЧС.
Для оценки специфичности метода использовали ДНК штамма «Ставрополь 01/08» вируса АЧС, а также ДНК гетерологичных вирусов, вызывающих болезни свиней с аналогичными клиническими признаками - ДНК штамма «Амурский-2019» вируса классической чумы свиней, ДНК штамма «Самара-96» вируса репродуктивно-респираторного синдрома свиней, ДНК штамма «Губкинский» вируса трансмиссивного гастроэнтерита свиней; ДНК штамма «Арский» вируса болезни Ауески из коллекции ФГБНУ ФИЦВиМ.
Петлевую изотермическую амплификацию проводили по программе с увеличенным количеством циклов для исключения ложноположительных реакций, время реакции составило 50 минут (стандартное рекомендуемое время исследования 30 минут) на амплификаторе CFX 96 (Bio-Rad) (таблица 2).
циклов
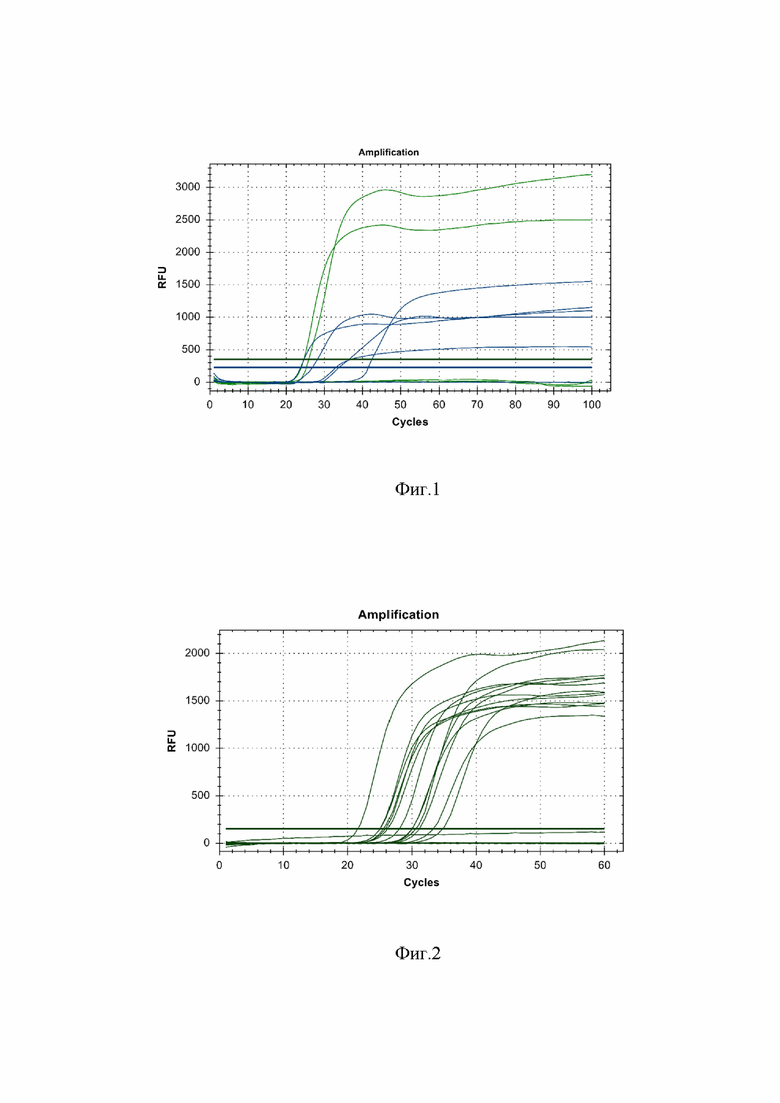
Полученные результаты представлены в таблице 3 и на фиг. 1.
Результаты интерпретировали, анализируя кривые накопления флуоресцентного сигнала для каждой пробы с помощью программного обеспечения прибора. Результаты интерпретировали на основании наличия или отсутствия значения Ct. Для образца содержащего ДНК штамма АЧС и положительного контроль амплификации был получен сигнал амплификации на 14 (Ct 25,92) и 13 (Ct 24,61) минуте исследования соответственно, в то время как образцы, содержащие ДНК других возбудителей, были отрицательные вплоть до 50 минут исследования.
ДНК вируса АЧС
Все образцы, содержащие ДНК гетерологичных вирусов, были определены как отрицательные, не содержащие ДНК вируса АЧС, при наличии сигнала выявления ДНК ВКО в диапазоне 14-22 минуты.
Пример 2. Определение чувствительности петлевой изотермической амплификации с гибридизационно-флуоресцентными зондами для выявления генома вируса АЧС.
Для определения аналитической чувствительности метода были приготовлены разведения ДНК рекомбинантной плазмиды, содержащей фрагмент генома вируса АЧС с известной концентрацией.
Петлевую изотермическую амплификацию проводили при 65°С в течение 30 минут с детекцией флуоресцентного сигнала каждые 30 сек на амплификаторе CFX 96 (Bio-Rad). Результаты представлены на фиг. 2 и в таблице 4.
Чувствительность предложенного метода составляет 2*10^3 копий/мл (20 копий ДНК на реакцию), ДНК вируса АЧС воспроизводимо выявляется во всех образцах, содержащих ДНК рекомбинантной плазмиды.
копий/мл
ДНК вируса АЧС
Пример 3. Определение диагностической чувствительности и специфичности петлевой изотермической амплификации с гибридизационно-флуоресцентными зондами.
Для испытаний диагностической чувствительности и специфичности LAMP методик использовали 42 образца плазмы крови и 21 образец суспензии тканей (миндалины, селезенка, легкие). Данные образцы были исследованы предварительно с использованием двух диагностических ПЦР наборов разных производителей с заявленной аналитической чувствительностью не менее 1*10^3 копий/мл, предназначенных для выявления вируса АЧС в биологическом материале от животных. Результаты выявления ДНК вируса АЧС ПЦР наборами дали полностью сопоставимые результаты, 22 из 42 образцов плазмы и 11 из 21 образца суспензий тканей содержали ДНК вируса АЧС (таблица 5).
Среди 33 образцов биологического материала, содержащего ДНК вируса АЧС, 26 образцов имели достаточную вирусную нагрузку с пороговыми циклами ПЦР Ctсред менее 35 циклов, 7 образцов имели низкое содержание ДНК вируса АЧС в образцах, ПЦР Ctсред более 35 циклов.
Петлевую изотермическую амплификацию проводили по программе с увеличенным количеством циклов для исключения ложноположительных реакций, время реакции составило 50 минут (стандартное рекомендуемое время исследования 30 минут) на амплификаторе CFX 96 (Bio-Rad) (таблица 2).
Способ представленный в данном патенте LAMP, позволяет обнаружить ДНК вируса АЧС во всех образцах с достаточной вирусной нагрузкой, время накопления флуоресценции до порогового уровня при LAMP составляет от 12 до 22 минут. Для образцов с низкой вирусной нагрузкой (ПЦР Ct сред> 35) метод LAMP позволил обнаружить вирус в 6 (7) образцов. Все 30 ПЦР отрицательные образцы были определены верно, даже при увеличении времени реакции до 50 мин, что указывает на высокую специфичность разработанного LAMP метода. Результаты представлены в таблице 5.
Ct
Ct
Ct сред.
Диагностические характеристики LAMP были рассчитаны с помощью интернет-ресурса MEDCALC [25] при внесении значений истинных (TP) и ложных (FP) положительных и истинных (TN) и ложных (FN) отрицательных результатов.
Было показано, что диагностическая чувствительность метода LAMP в реальном времени с флуоресцентными зондами практически не уступает по чувствительности ПЦР и составляет 97,0 %. При этом наблюдается диагностическая специфичность до 100%, все образцы, не содержащие ДНК вируса АЧС определены верно (табл 6).
Литература:
1. Chapman DAG, Darby AC, Da Silva M, Upton C, Radford AD, Dixon LK. Genomic analysis of highly virulent isolate of African swine fever virus. Emerg Infect Dis. 2011 Apr.
2. Blome S, Gabriel C, Beer M. Pathogenesis of African swine fever in domestic pigs and European wild boar. Virus Res. 2013 Apr; 173(1):122-30. doi: 10.1016/j.virusres.2012.10.026. Epub 2012 Nov 6. PMID: 23137735.
3. Dixon LK, Stahl K, Jori F, Vial L, Pfeiffer DU. African Swine Fever Epidemiology and Control. Annu Rev Anim Biosci. 2020 Feb 15;8:221-246. doi: 10.1146/annurev-animal-021419-083741. Epub 2020 Nov 19. PMID: 31743062.
4. Manual of Diagnostic Tests and Vaccines for Terrestrial Animals 2022 , World Organization for Animal Health, 2022.
5. Бельтран Алькрудо, Д., Ариас, М., Гайардо, К., Крамер, С. и Пенрит, М.Л Африканская чума свиней: обнаружение и диагностика - Руководство для ветеринаров, ФАО, 2017. Руководство по животноводству и охране здоровья животных № 19. Рим. Продовольственная и сельскохозяйственная организация Организации Объединенных Наций (ФАО). 104 стр.
6. Notomi T, Okayama H, Masubuchi H, Yonekawa T, Watanabe K, Amino N, Hase T. Loop-mediated isothermal amplification of DNA. Nucleic Acids Res. 2000 Jun 15;28(12): E63. doi: 10.1093/nar/28.12.e63. PMID: 10871386; PMCID: PMC102748.
7. Nagamine K, Hase T, Notomi T. Accelerated reaction by loop-mediated isothermal amplification using loop primers. Mol Cell Probes. 2002 Jun;16(3):223-9. doi: 10.1006/mcpr.2002.0415. PMID: 12144774.
8. Mori Y, Nagamine K, Tomita N, Notomi T. Detection of loop-mediated isothermal amplification reaction by turbidity derived from magnesium pyrophosphate formation. Biochem Biophys Res Commun. 2001 Nov 23;289(1):150-4. doi: 10.1006/bbrc.2001.5921. PMID: 11708792.
9. Tanner NA, Zhang Y, Evans TC Jr. Visual detection of isothermal nucleic acid amplification using pH-sensitive dyes. Biotechniques. 2015 Feb 1;58(2):59-68. doi: 10.2144/000114253. PMID: 25652028.
10. Wang D, Yu J, Wang Y, Zhang M, Li P, Liu M, Liu Y. Development of a real-time loop-mediated isothermal amplification (LAMP) assay and visual LAMP assay for detection of African swine fever virus (ASFV). J Virol Methods. 2020 Feb;276:113775. doi: 10.1016/j.jviromet.2019.113775. Epub 2019 Nov 11. PMID: 31726114.
11. Wang Y, Dai J, Liu Y, Yang J, Hou Q, Ou Y, Ding Y, Ma B, Chen H, Li M, Sun Y, Zheng H, Zhang K, Wubshet AK, Zaberezhny AD, Aliper TI, Tarasiuk K, Pejsak Z, Liu Z, Zhang Y, Zhang J. Development of a Potential Penside Colorimetric LAMP Assay Using Neutral Red for Detection of African Swine Fever Virus. Front Microbiol. 2021 Apr 23;12:609821. doi: 10.3389/fmicb.2021.609821. PMID: 33967972; PMCID: PMC8102904.
12. - Miao, F.; Zhang, J.; Li, N.; Chen, T.; Wang, L.; Zhang, F.; Mi, L.; Zhang, J.; Wang, S.; Wang, Y.; et al. Rapid and Sensitive Recombinase Polymerase Amplification Combined with Lateral Flow Strip for Detecting African Swine Fever Virus. Front. Microbiol. 2019, 10, 1004.
13.- Yu LS, Chou SY, Wu HY, Chen YC, Chen YH. Rapid and semi-quantitative colorimetric loop-mediated isothermal amplification detection of ASFV via HSV color model transformation. J Microbiol Immunol Infect. 2021 Oct;54(5):963-970. doi: 10.1016/j.jmii.2020.08.003. Epub 2020 Aug 16. PMID: 32868194.
14. Kubota R, Vine BG, Alvarez AM, Jenkins DM. Detection of Ralstonia solanacearum by loop-mediated isothermal amplification. Phytopathology. 2008 Sep;98(9):1045-51. doi: 10.1094/PHYTO-98-9-1045. PMID: 18943743.
15. Tyagi S Kramer F. 1996. Molecular Beacons: Probes That Fluoresce Upon Hybridization. Nature biotechnology. 14. 303- 8. 10.1038/nbt0396- 303).
6. Heid CA, Stevens J, Livak KJ, and Williams PM (1996) Real time quantitative PCR. Genome Res 6(10): 986-994.
17. - Zsak, L., Borca, M. V., Risatti, G. R., Zsak, A., French, R. A., Lu, Z., …Rock, D. L. (2005). Preclinical diagnosis of African swine fever in contact-exposed swine by a real-time PCR assay. Journal of Clinical Microbiology, 43, 112-119. https://doi.org/10.1128/JCM.43.1.112-119.2005.
18. Wang Y, Xu L, Noll L, Stoy C, Porter E, Fu J, Feng Y, Peddireddi L, Liu X, Dodd KA, Jia W, Bai J. Development of a real-time PCR assay for detection of African swine fever virus with an endogenous internal control. Transbound Emerg Dis. 2020 Nov;67(6):2446-2454. doi: 10.1111/tbed.13582. Epub 2020 May 2. PMID: 32306531.
19. Tignon M, Gallardo C, Iscaro C, Hutet E, Van der Stede Y, Kolbasov D, De Mia GM, Le Potier MF, Bishop RP, Arias M, Koenen F. Development and inter-laboratory validation study of an improved new real-time PCR assay with internal control for detection and laboratory diagnosis of African swine fever virus. J Virol Methods. 2011 Dec;178(1-2):161-70. doi: 10.1016/j.jviromet.2011.09.007. Epub 2011 Sep 17. PMID: 21946285.
20.  -Pinero J, Gallardo C, Elizalde M, Robles A,
-Pinero J, Gallardo C, Elizalde M, Robles A,  C, Bishop R, Heath L, Couacy-Hymann E, Fasina FO, Pelayo V, Soler A, Arias M. Molecular diagnosis of African Swine Fever by a new real-time PCR using universal probe library. Transbound Emerg Dis. 2013 Feb;60(1):48-58. doi: 10.1111/j.1865-1682.2012.01317.x. Epub 2012 Mar 7. PMID: 22394449.
C, Bishop R, Heath L, Couacy-Hymann E, Fasina FO, Pelayo V, Soler A, Arias M. Molecular diagnosis of African Swine Fever by a new real-time PCR using universal probe library. Transbound Emerg Dis. 2013 Feb;60(1):48-58. doi: 10.1111/j.1865-1682.2012.01317.x. Epub 2012 Mar 7. PMID: 22394449.
21. Zhao, D. , Liu, R. , Zhang, X. , Li, F. , Wang, J. , Zhang, J. , Liu, X. , Wang, L. , Zhang, J. , Wu, X. , Guan, Y. , Chen, W. , Wang, X. , He, X. , & Bu, Z. (2019). Replication and virulence in pigs of the first African swine fever virus isolated in China. Emerging Microbes & Infections, 8(1), 438-447.
22. National Center for Biotechnology Information (NCBI) [Электронный ресурс] URL: https://www.ncbi.nlm.nih.gov.
23. Oligo Calc: Oligonucleotide Properties Calculator [Электронный ресурс] URL: http:// biotools.nubic.northwestern.edu/ OligoCalc. Html
24. Development of multiplexed reverse-transcription loop-mediated isothermal amplification for detection of SARS-CoV-2 and influenza viral RNA Yinhua Zhang and Nathan A Tanner, BioTechniques 2021 70:3, 167-174.
25. MedCalc's Diagnostic test evaluation calculator https://www.medcalc.org/calc/diagnostic-test.php
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="СИНТЕТИЧЕСКИЕ
ОЛИГОНУКЛЕОТИДНЫЕ.xml" softwareName="WIPO Sequence"
softwareVersion="2.1.2" productionDate="2022-09-30">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText></ApplicationNumberText>
<FilingDate></FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>01-09-22</ApplicantFileReference>
<ApplicantName languageCode="ru">Федеральное государственное
бюджетное образовательное учреждение высшего образования «Московская
государственная академия ветеринарной медицины и биотехнологии – МВА
имени К.И. Скрябина» (ФГБОУ ВО МГАВМиБ - МВА имени К.И.
Скрябина)</ApplicantName>
<ApplicantNameLatin>Federalnoe gosudarstvennoe biudzhetnoe
obrazovatelnoe uchrezhdenie vysshego obrazovaniia "Moskovskaia
gosudarstvennaia akademiia veterinarnoi meditsiny i biotekhnologii
MVA imeni K.I. Skriabina, FGBOU VO MGAVMiB - MVA imeni K.I.
Skriabina</ApplicantNameLatin>
<InventionTitle languageCode="ru">СИНТЕТИЧЕСКИЕ ОЛИГОНУКЛЕОТИДНЫЕ
ПРАЙМЕРЫ И СПОСОБ ВЫСОКОЧУВСТВИТЕЛЬНОГО ЭКСПРЕСС ВЫЯВЛЕНИЯ ДНК ВИРУСА
АФРИКАНСКОЙ ЧУМЫ СВИНЕЙ МЕТОДОМ ПЕТЛЕВОЙ ИЗОТЕРМИЧЕСКОЙ АМПЛИФИКАЦИИ
В ПРИСУТСТВИИ ДНК ВНУТРЕННЕГО КОНТРОЛЬНОГО ОБРАЗЦА</InventionTitle>
<SequenceTotalQuantity>16</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>African swine fever
virus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>attacgtcttatgtccaga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>African swine fever
virus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agactggatataagcacttg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q3">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>African swine fever
virus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcacaatatccaaacagcaggt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>African swine fever
virus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>taagaataggtttgctytggtg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>47</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..47</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q5">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>African swine fever
virus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>accaacccgaaattcctttcactttttgcgtccgtaatagrgtrata</
INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>African swine fever
virus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gttcgctcgtatcattttcatttttaggtatcggtggagggaacy</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q7">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>African swine fever
virus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aaacgcaacgaggctcta</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>African swine fever
virus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggatttgttcagaacgctc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q9">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>African swine fever
virus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>acagcttccgctgtcttctca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>African swine fever
virus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gttgacgacgacatggctc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>41</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..41</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q11">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>African swine fever
virus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cctgctgatctgcgacttatttcgagagtgcgttgcttaac</INSDSe
q_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>44</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..44</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>African swine fever
virus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggattccaaagttctcaatgcttttgagaatcgcagcaacttgt</INS
DSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="13">
<INSDSeq>
<INSDSeq_length>47</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..47</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q13">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>African swine fever
virus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>accaacccgaaattcctttcactttttgcgtccgtaatagrgtrata</
INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="14">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>African swine fever
virus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtgaaaggaatttcgggttggt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="15">
<INSDSeq>
<INSDSeq_length>41</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..41</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q15">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>African swine fever
virus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cctgctgatctgcgacttatttcgagagtgcgttgcttaac</INSDSe
q_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="16">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q16">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>African swine fever
virus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ataagtcgcagatcagcagg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
Изобретение относится к области молекулярной диагностики. Описаны набор олигонуклеотидных праймеров и зонды для выявления ДНК вируса африканской чумы свиней (АЧС) и ДНК бактериофага лямбда как внутреннего контрольного образца исследования. Также предложен способ выявления ДНК вируса АЧС из биологического материала с использованием набора олигонуклеотидных праймеров и зондов с помощью петлевой изотермической амплификации (LAMP) в присутствии внутреннего контрольного образца ДНК бактериофага лямбда. Технический результат заключается в разработке высокочувствительного, специфичного и быстрого метода выявления вируса африканской чумы свиней. 2 н. и 1 з.п. ф-лы, 2 ил., 6 табл., 3 пр.
1. Набор синтетических олигонуклеотидных праймеров для выявления ДНК вируса африканской чумы свиней методом петлевой изотермической амплификации в режиме реального времени, содержащий олигонуклеотидные праймеры ASF-F3, ASF-В3, ASF-F, ASF-BL, ASF-FIP, ASF-BIP, фланкирующие специфический фрагмент генома вируса АЧС, олигонуклеотидные праймеры IC-F3, IC-В3, IC-F, IC-BL, IC-FIP, IC-BIP, фланкирующие специфический фрагмент бактериофага лямбда, а также зонды ASF-FIP-BHQ1 / ASF-R6G, IC-FIP-BHQ1 / IC-FAM на основе внутренних праймеров FIP, имеющие следующий нуклеотидный состав:
ASF-F3 ATTACGTCTTATGTCCAGA,
ASF-В3 AGACTGGATATAAGCACTTG,
ASF-F TCACAATATCCAAACAGCAGGT,
ASF-BL TAAGAATAGGTTTGCTYTGGTG,
ASF-FIP ACCAACCCGAAATTCCTTTCACTTTTTGCGTCCGTAATAGRGTRATA,
ASF-BIP GTTCGCTCGTATCATTTTCATTTTTAGGTATCGGTGGAGGGAACY,
IC-F3 AAACGCAACGAGGCTCTA,
IC-В3 GGATTTGTTCAGAACGCTC,
IC-FL ACAGCTTCCGCTGTCTTCTCA,
IC-BL GTTGACGACGACATGGCTC,
IC-FIP CCTGCTGATCTGCGACTTATTTCGAGAGTGCGTTGCTTAAC,
IC -BIP GGATTCCAAAGTTCTCAATGCTTTTGAGAATCGCAGCAACTTGT
ASF-FIP-BHQ1 / ASF-R6G
BHQ1-ACCAACCCGAAATTCCTTTCACTTTTTGCGTCCGTAATAGRGTRATA,
GTGAAAGGAATTTCGGGTTGGT-R6G,
IC-FIP-BHQ1 / IC- FAM
BHQ1-CCTGCTGATCTGCGACTTATTTCGAGAGTGCGTTGCTTAAC,
ATAAGTCGCAGATCAGCAGG-FAM.
2. Способ высокочувствительного экспресс-выявления ДНК вируса африканской чумы свиней методом петлевой изотермической амплификации в присутствии ДНК внутреннего контрольного образца, включающий проведение амплификации ДНК вируса АЧС, выделенной из биологического материала от животных в режиме «реального времени» изотермическим способом с использованием набора олигонуклеотидных праймеров по п. 1, в составе реакционной 2,5 x смеси LAMP, содержащей праймеры в следующих концентрациях ASF-FIP, IC-FIP - по 1.0 μM, ASF-BIP, IC-BIP - по 2.5 μM, ASF-F3, ASF-B3, IC-F3, IC-B3 - по 0.5 μM, ASF-FL, IC-FL, ASF-BL, IC-BL - 1.0 μM, зондов-праймеров ASF-FIP-BHQ1, IC-FIP-BHQ1 по 1.5 μM, ASF-R6G , IC-FAM - 2.0 μM, 1,4 мМ каждого дНТФ после предварительной экстракции ДНК из исследуемых проб в присутствии бактериофага лямбда, добавляемого в каждый образец до концентрации в образце 10^5–10^6 копий/мл, с использованием для проведения LAMP положительных и отрицательных контрольных образцов этапов экстракции и амплификации ДНК, объем подготовленной смеси для одной пробы составляет 15 мкл, из них 10 мкл реакционная 2,5 x смесь LAMP; 5 мкл 5 x LAMP буфера; 1 мкл Bst ДНК полимераза с активностью 8 ед./мкл, компоненты реакционной смеси перемешивают и вносят в подготовленные для амплификации пробирки по 15 мкл, затем в пробирки вносят по 10 мкл проб ДНК, включая контрольные реакции, программируют амплификатор с системой флуоресцентной детекции для выполнения следующей программы – 65 °С, 30 с, детекция после каждого цикла на каналах FAM/Green, HEX/Yellow, устанавливают 60 циклов, по завершении амплификации проводят анализ полученных данных, анализируют кривые накопления флуоресцентного сигнала, результаты интерпретируют на основании наличия или отсутствия пересечения кривой флуоресценции с установленной на соответствующем уровне пороговой линией, соответствующем 10-20 % от максимального уровня флуоресценции, полученного для образца К+ в последнем цикле амплификации, интерпретация результатов следующая - обнаружена ДНК вируса АЧС, если определено значение Ct на канале для флуорофора HEX/Yellow, не обнаружена ДНК вируса АЧС, если отсутствует значение Ct на канале для флуорофора HEX/Yellow, при этом значение Сt по каналу для флуорофора FAM/Green определено менее граничного значения, Сt ≤45, если отсутствует значение Ct на канале для флуорофора HEX/Yellow, при этом, если значение Сt по каналу для флуорофора FAM/Green не определено, проводят повторное исследование образца, результаты подлежат учету, только если получены корректные результаты для контролей этапов экстракции и амплификации ДНК.
3. Способ по п. 2, характеризующийся тем, что может включать проведение амплификации ДНК вируса АЧС с использованием праймеров по п. 1 в других соотношениях и концентрациях.
| Тест-система для обнаружения ДНК вируса африканской чумы свиней с помощью полимеразной цепной реакции в режиме реального времени | 2017 |
|
RU2645263C1 |
| Синтетические олигонуклеотидные праймеры и способ выявления ДНК вируса АЧС методом петлевой изотермической амплификации | 2018 |
|
RU2710065C1 |
| CN 110453010 A, 15.11.2019 | |||
| Aguero M | |||
| et al | |||
| Highly sensitive PCR assay for routine diagnosis of African swine fever virus in clinical samples //Journal of clinical microbiology | |||
| Способ и приспособление для нагревания хлебопекарных камер | 1923 |
|
SU2003A1 |
| - Vol | |||
| Механический грохот | 1922 |
|
SU41A1 |
| - n | |||
| Разборный с внутренней печью кипятильник | 1922 |
|
SU9A1 |
| - P | |||
| Устройство для контролирования состояния заказов в производстве | 1926 |
|
SU4431A1 |
Авторы
Даты
2023-07-05—Публикация
2022-10-06—Подача