Изобретение относится к областям биотехнологии и медицины, а именно к молекулярной онкологии, и касается способа выявления специфического химерного транскрипта DNAJB1-PRKACA в образцах отдельного подтипа гепатоцеллюлярной карциномы (ГЦК) - фиброламеллярной карциномы (ФлК) (Шавочкина Д.А., Кустова И.Ф., Лазаревич Н.Л. Фиброламеллярная карцинома как отдельный подтип гепатоцеллюлярного рака: молекулярно-генетические особенности, диагностика, перспективы лечения // Злокачественные опухоли.- 2017. - N 3 (7). - С. 71-80), которая возникает обычно у молодых пациентов и характеризуется особым морфологическим строением. Предложенный способ может быть использован в клинической практике для надежной дифференциальной диагностики фиброламеллярной карциномы от других форм опухолей печени для выбора оптимальной тактики лечения специфического заболевания (O'Neill A.F., Church A.J., Perez-Atayde A.R., Shaikh R., Marcus K.J., Vakili K. Fibrolamellar carcinoma: An entity all its own // Current Problems in Cancer. - 2021. - Vol. 45, No. 4. - P. 100770).
В 2014 году при проведении полнотранскриптомного анализа образцов ФлК был открыт химерный транскрипт DNAJB1-PRKACA, который образуется в результате делеции участка 19 хромосомы размером 400 тысяч пар нуклеотидов (т.п.н.), приводящей к слиянию двух генов (Honeyman J.N., Simon Е.Р. Robine N., Chiaroni-Clarke R., Darcy D.G., Lim LLP., Gleason C.G., Murphy J., Rosenberg B.R., Teegan L., Takacs C.N., Botero S., Belote R., Germer S., Emde A-K., Vacic V., Bhanot U., Michael P. LaQuaglia M.P., Simon S.F. Detection of a Recurrent DNAJB1-PRKACA Chimeric Transcript in Fibrolamellar Hepatocellular Carcinoma // Science. - 2014. - Vol. 343, No. 6174. - P. 1010-1022). Эта перестройка изменяет биологические свойства генов, формирующих химерный транскрипт, и, как следствие, белков, кодируемых этими генами. Образование химерного транскрипта DNAJB1-PRKACA специфично для ФлК, является ключевым генетическим нарушением при формировании этой формы опухолей и рассматривается в качестве перспективной мишени для разработки таргетных препаратов для терапии этой формы опухолей. В настоящее время в клинической практике дифференциальная диагностика ФлК от других форм опухолей печени осуществляется на основании гистологического исследования постоперационных срезов, однако этот способ является времязатратным, требует значительной квалификации исполнителя и не всегда позволяет получить однозначный ответ.
В международной заявке «Method for detecting DNAJB1-PRKACA gene», описывающей варианты методов выявления химерного транскрипта или кодируемых им белковых последовательностей, был представлен специфический набор праймеров для выявления транскрипта DNAJB1-PRKACA с помощью различных молекулярно-биологических методов (WO 2015/129655 А1).
Недостатком этого метода является невозможность верифицировать запатентованный набор праймеров. Данных об использовании референсного гена в заявке не содержится.
В 2015 году в научной статье Graham и соавторами было предложено 3 метода для выявления хромосомной перестройки DNAJB1-PRKACA или образующегося в ее результате химерного транскрипта (Graham R.P., Jin L., Knutson D.L., Kloft-Nelson S.M., Greipp P.T., Waldburger N., Roessler S., Longerich Т., Roberts L.R., Oliveira A.M., Kevin С Hailing K.C., Schirmacher P., Torbenson M.S. DNAJB1-PRKACA is specific for fibrolamellar carcinoma // Modern Pathology. - 2015. - Vol. 28, No. 6. - P. 822-829).
1. Метод флуоресцентной гибридизации in situ (FISH), применяемый для диагностики хромосомных аномалий. Метод основан на использовании ДНК-проб, комплементарных исследуемому участку ДНК, со специфической флуоресцентной меткой.
R.P. Graham и соавторы использовали FISH метод для выявления образующейся в результате хромосомной перестройки последовательности ДНК слитного гена DNAJB1-PRKACA на парафиновых срезах образцов опухолей печени человека.
Недостатком этого метода являются трудоемкость, потребность в специализированных реактивах и оборудовании, высокая стоимость и времязатратность.
2. Метод РНК гибридизации in situ (RNA ISH), позволяющий полуколичественно оценить экспрессию РНК на парафиновых срезах исследуемых образцов ткани. Graham и соавторы использовали этот метод для оценки уровня экспрессии химерного транскрипта DNAJB1-PRKACA и неизмененного транскрипта PRKACA с помощью специфических олигонуклеотидных зондов.
К недостаткам этого метода относится высокая трудоемкость, субъективность оценки результатов анализа и высокая стоимость используемых специализированных реактивов.
3. Метод полимеразной цепной реакции (ПЦР) с обратной транскрипцией с детекцией по конечной точке со специфическими праймерами к химерному транскрипту DNAJB1-PRKACA, предложенный в 2015 году R.P. Graham и соавторами, является прототипом заявляемого способа. В качестве объекта исследования в этом методе используются препараты кДНК, полученные в результате обратной транскрипции образцов тотальной РНК, выделенной из парафиновых срезов, фиксированных в формалине опухолевых тканей после проведения микродиссекции, в количестве, эквивалентном 50-100 нг РНК на одну реакцию.
Дизайн праймеров, используемых для ПЦР, проведен таким образом, что левый праймер соответствует последовательности экзона 1 гена DNAJB1, а правый - экзона 2 гена PRKACA. В результате ПЦР происходит амплификация только химерных транскриптов, содержащих последовательности, комплементарные последовательностям обоих генов, что обеспечивает специфичность реакции. Для проверки наличия и качества взятой кДНК используется независимая реакция с праймерами к референсному гену фосфоглицеролкиназы 1 (PGK1). Метод не является количественным, по окончании реакции его результат для каждого из двух транскриптов анализируют путем электрофореза продуктов ПЦР в 3% агарозном геле, окрашиваемом бромистым этидием, по наличию/отсутствию продукта ожидаемого размера.
Недостатком метода является его трудоемкость, времязатратность, необходимость использования значительных количеств образцов РНК, невысокая чувствительность детекции химерного транскрипта и, как следствие, возможность получения ложно-отрицательных результатов, невозможность количественной оценки результатов независимой амплификации двух разных транскриптов в разных реакциях.
Задачей изобретения является разработка нового максимально чувствительного и унифицированного метода детекции химерного транскрипта DNAJB1-PRKACA и оценки его относительного количества в клинических образцах фиброламеллярной карциномы печени человека.
Технический результат изобретения заключается в том, что заявляемый способ. обладает высокой специфичностью и чувствительностью, позволяет с высокой достоверностью детектировать химерный транскрипт DNAJB1-PRKACA в исследуемых образцах. Изобретение является доступным для применения в клинической практике.
Технический результат достигается тем, что проводят ПЦР в режиме реального времени со специфическими олигонуклеотидными последовательностями - праймерами (SEQ ID NO 1, NO 2; SEQ ID NO 4, NO 5) и двумя специально адаптированными флуоресцентными зондами (SEQ ID NO 3, ID NO 6) для повышения чувствительности способа.
Для реализации технического результата используют препараты кДНК, полученные в результате реакции обратной транскрипции со случайными гексамерными праймерами образцов обработанной ДНКазой I суммарной РНК, выделенной из различных типов (свежие, замороженные в жидком азоте или в консервирующем растворе (RNALater или аналоги) образцов опухолевой и (опционально, в качестве отрицательного контроля) неопухолевой ткани печени, полученных при резекции опухолей, либо аналогичным образом законсервированного биопсийного материала. При необходимости могут быть также использованы образцы РНК, выделенной из парафиновых срезов, фиксированных в формалине опухолевой ткани, однако в этом случае количество РНК и ее целостность будут значимо снижены.
Полученные препараты кДНК используют в качестве матрицы в мультиплексной ПЦР в реальном времени со специфическими праймерами и флуоресцентными зондами TaqMan (линейные разрушаемые зонды), мечеными по 5'-концу флуорофором, а по 3'-концу - гасителем флуоресценции. Детекция результатов реакции осуществляется на каждом цикле амплификации за счет флуоресценции TaqMan-зонда, которая линейно возрастает за счет его разрушения полимеразой при связывании со специфической последовательностью внутри анализируемого ампликона.
Отличительным признаком заявляемого изобретения является система из четырех праймеров и двух флуоресцентных зондов, которая позволяет специфично детектировать в одной реакции маркерный для ФлК химерный транскрипт DNAJB1-PRKACA и транскрипт референсного гена С-концевого связывающего белка 1 (СТВР1), который равномерно экспрессируется в нормальных тканях печени и в ГЦК различной этиологии и используется для контроля количества и целостности препаратов РНК, использованных для анализа.
Последовательности олигонуклеотидов приведены в Перечне последовательностей.
Копия Перечня последовательностей олигонуклеотидов, представленная на машиночитаемом носителе, идентична Перечню последовательностей олигонуклеотидов в печатной форме.
Используются следующие последовательности олигонуклеотидов:
1) Праймеры:
а. DNAJB1-PRKACA
i. Левый TTCAAGGAGATCGCTGAGGC (SEQ ID NO 1)
ii. Правый TGTGTTCTGAGCGGGACTTT (SEQ ID NO 2)
б. CTBP1
iii. Левый AAGGATGCACCCAACCTCAT (SEQ ID NO 4)
iv. Правый GATGGTCCTTGTTGACACAGT (SEQ ID NO 5)
2) Флуоресцентно меченые TaqMan зонды:
а. DNAJB1-PRKACA HEX-CGCTACGGGGAGGAAGTGAA-BHQ1 (SEQ. ID NO 3)
б. CTBP1 FAM-CGAGAGGAGGCGGCACGGGA-BHQ1 (SEQ ID NO 6)
Подбор последовательностей праймеров и зондов включает дизайн олигонуклеотидов, строго комплементарных целевым последовательностям выявляемых транскриптов. Длина амплифицируемого участка составляет 147 пар нуклеотидов (п.н.) для слитного транскрипта DNAJB1-PRKACA и 154 п.н. для СТВР1. Дизайн праймеров, используемых для ПЦР химерного транскрипта, проведен таким образом, что левый праймер соответствует последовательности экзона 1 гена DNAJB1, а правый лежит на стыке 2 и 3 экзонов гена PRKACA, что обеспечивает специфическую амплификацию только химерных транскриптов, содержащих последовательности, комплементарные последовательностям обоих генов. Дополнительное повышение специфичности реакции достигается использованием флуоресцентного зонда, комплементарного последовательностям, лежащим на стыке двух генов.
Амплификацию проводят в режиме реального времени на приборах, обеспечивающих возможность детектировать флуоресценцию в двух каналах: FAM и HEX, например, CFX Connect, CFX 96 или CFX Opus Real-Time PCR Detection System (Bio-Rad).
Состав ПЦР-смеси для постановки одной реакции (25 мкл) включает:
- по 0,2 пкмоль/мкл каждого из 4 праймеров,
- по 0,1 пкмоль/мкл каждого из двух флуоресцентных зондов,
- кДНК исследуемого образца (здесь и далее: условные нг - количество кДНК, получаемое при обратной транскрипции 1 нг суммарной РНК, у.нг) - 15 (допустимый интервал концентраций - 0,5-32) у.нг;
а также компоненты набора для проведения ПЦР в реальном времени (например, Синтол, каталожный номер R-412):
- dNTP с конечной концентрацией в реакции 0,25 мМ,
- ПЦР буфер без MgCl2 с конечной концентрацией в реакции 1х,
- MgCl2 с конечной концентрацией в реакции 2,5 мМ,
- SynTaq ДНК-полимераза с конечным количеством в реакции 1,5 единиц активности.
Режим проведения ПЦР:
1 этап: денатурация при t=95°C в течение 5 мин
2 этап: 40 циклов ПЦР:
- денатурация при t=95°С в течение 30 сек
- отжиг олигонуклеотидов при t=62°С в течение 30 сек
- элонгация при t=72°С в течение 50 сек
- детекция флуоресцентного сигнала
3 этап: элонгация при t=72° в течение 5 мин.
Детекцию флуоресцентного сигнала проводят на каждом цикле второго этапа немедленно после завершения элонгации.
Реакцию проводят со следующим набором проб:
• Вода (проба, в которую вместо исследуемого образца вносится вода, на которой готовится вся реакция)
• Контрольный образец РНК без ревертирования (проба, в которой в качестве матрицы используется РНК опухоли ФлК, подвергнутая реакции ревертирования без добавления фермента обратной транскриптазы)
• Исследуемые пробы - образцы опухолевой и (опционально) неопухолевой ткани печени того же пациента.
• При необходимости проведения количественной оттенки (см. ниже) -калибровочная кривая из трех последовательных четырехкратных разведений положительного по наличию химерного транскрипта DNAJB1-PRKACA образца кДНК (из опухоли ФлК), например 32, 8 и 2 у.нг кДНК. У.нг кДНК равны массе РНК, внесенной в реакцию ревертирования, выраженной в нг системы СИ.
Постановку каждой реакции проводят в трех технических повторах: для каждого повтора смешивают отдельный мастер микс, исследуемый образец кДНК разводят до нужной концентрации один раз для всех трех повторов.
Интерпретация результатов ПЦР.
Интерпретация результатов мультиплексной ПЦР в реальном времени проводят с использованием встроенного программного обеспечения для используемого амплификатора (например, Bio-Rad CFX Maestro) согласно рекомендациям производителя.
Реакция считается положительной, если сигнал регистрируется не менее, чем в 2 из трех технических реплик, при этом:
• Ct (среднее значение порогового цикла между техническими повторностями) референсного транскрипта СТВР1 должен быть меньше либо равен 36, в. противном случае необходимо переставить ПЦР с большим количеством образца или повторить исследование со свежим препаратом РНК.
• Химерный транскрипт считается экспрессирующимся, если в реакции Ct сигнала соответствующего зонда меньше либо равен 36.
• В контрольных реакциях с водой и контролем без ревертирования сигнал не детектируется до 38 цикла.
Изобретение иллюстрируется фигурами 1 и 2.
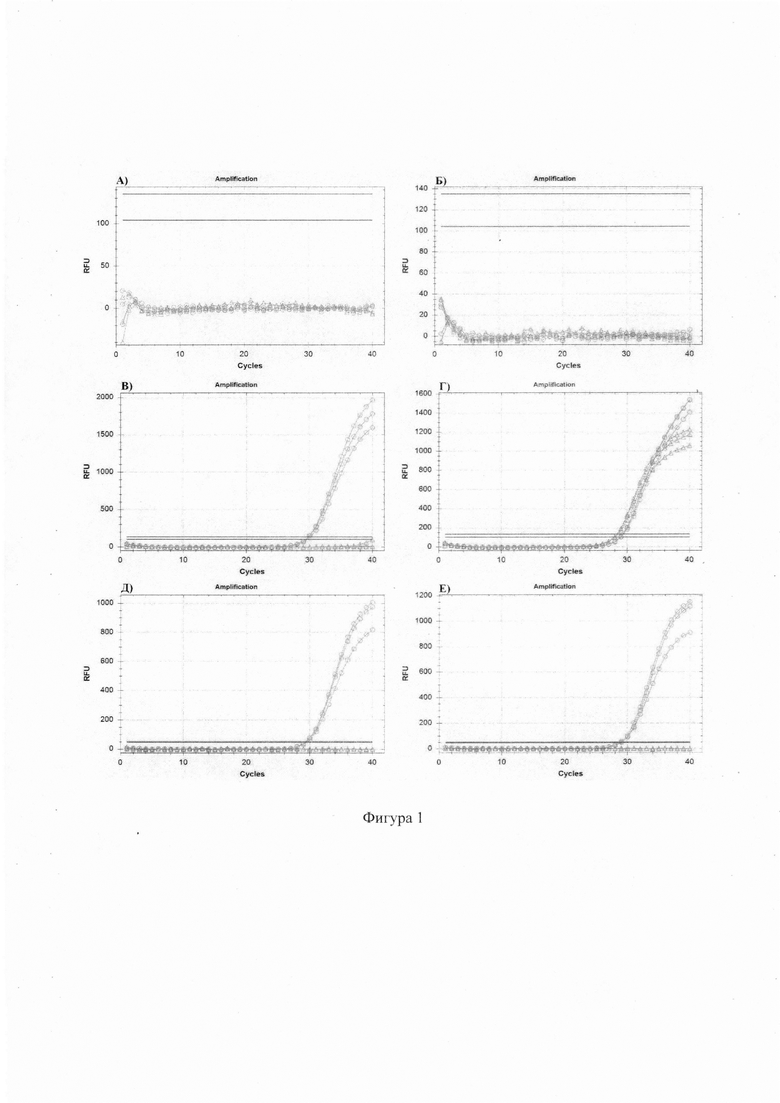
Фигура 1. Результаты мультиплексной реакции с праймерами и зондами на DNAJB1-PRKACA (канал HEX) и СТВР1 (канал FAM) в образцах кДНК из опухолевой (О) и неопухолевой (П) ткани печени. A) NTC - образец - вода. Б) NRT - образец - 20 нг РНК ФлК ГЦК 144-0 без ревертирования, В) Матрица - 15 у.нг кДНК ФлК ГЦК 087-П, Г) матрица - 15 у.нг кДНК ФлК ГЦК 087-О, Д) матрица - 15 у.нг кДНК нефиброламеллярной ГЦК 83-П, Е) матрица - 15 у.нг кДНК нефиброламеллярной ГЦК 83 О. Кружками отмечен сигнал FAM, треугольниками - HEX.
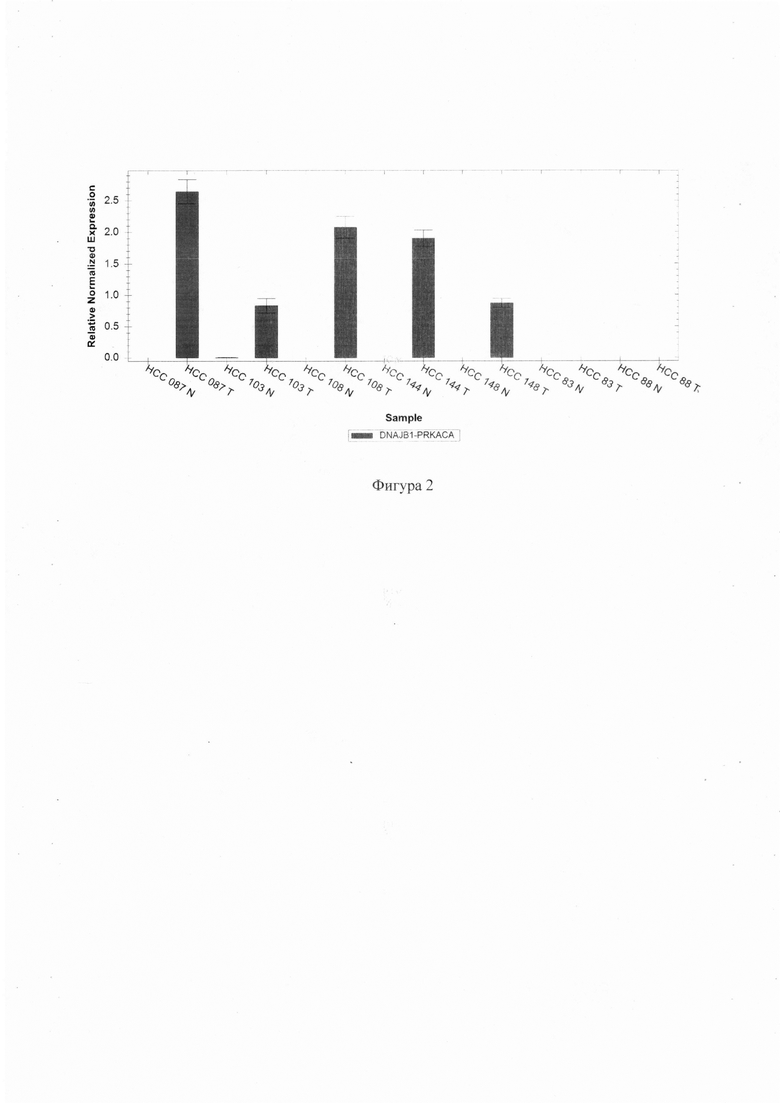
Фигура 2. Относительный уровень экспрессии химерного транскрипта DNAJB1-PRKACA, нормализованный по экспрессии референсного гена СТВР1, для 7 пар образцов неопухолевой (N) и опухолевой (Т) ткани печени 5 случаев ФлК (НСС 087, 103, 108, 144, 148) и 2 случаев нефиброламеллярной (НСС 83, 88) формы ГЦК. Анализ результатов ПЦР проведен с использованием программы Bio-Rad CFX Maestro в режиме нормализованной экспрессии (ΔΔCq).
Пример полученных результатов представлен на фигуре 1. На ней приведены результаты мультиплексной реакции с праймерами и зондами на DNAJB1-PRKACA (канал HEX) и СТВР1 (канал FAM) в образцах кДНК из опухолевой (О) и неопухолевой (П) ткани печени. A) NTC - образец - вода. Б) NRT - образец - 20 нг РНК ФлК ГЦК 144-0 без ревертирования, В) Матрица - 15 у.нг кДНК ФлК ГЦК 087-П, Г) матрица - 15 у.нг кДНК ФлК ГЦК 087-О, Д) матрица - 15 у.нг кДНК нефиброламеллярной ГЦК 83-П, Е) матрица - 15 у.нг кДНК нефиброламеллярной ГЦК 83 О. Зелеными кружками отмечен сигнал FAM, красными треугольниками - HEX.
При наличии калибровочной кривой с использованием положительного контрольного образца может быть проведен количественный анализ полученных результатов. В этом случае по окончании реакции в соответствующей программе (например, Bio-Rad CFX Maestro) на основании калибровочной кривой для каждого канала проводится определение значения порогового цикла Ct и условного количества продукта для ампликонов DNAJB1-PRKACA и СТВР1, а также определяется относительное количество химерного транскрипта относительно референсного в каждом из образцов. Пример результатов количественного анализа для 7 пар образцов различных форм ГЦК и неопухолевой ткани печени представлен на фигуре 2.
Изобретение иллюстрируется примерами 1-7, представляющими исследование парных образцов опухолевой и неопухолевой ткани печени при различных формах ГЦК.
Пример 1.
Идентификатор: ГЦК 83
Возраст: 74 года
Диагноз: нефиброламеллярная форма ГЦК.
Выполнен анализ наличия экспрессии DNAJB1-PRKACA заявленным способом.
Результат: При исследовании иссеченного тканевого материала пациента экспрессия DNAJB1-PRKACA не обнаружена ни в ткани опухоли, ни в прилежащей условно здоровой ткани печени.
Пример 2.
Идентификатор: ГЦК 88
Возраст: 53 года
Диагноз: нефиброламеллярная форма ГЦК.
Выполнен анализ наличия экспрессии DNAJB1-PRKACA заявленным способом.
Результат: При исследовании иссеченного тканевого материала пациента экспрессия DNAJB1-PRKACA не обнаружена ни в ткани опухоли, ни в прилежащей условно здоровой ткани печени.
Пример 3.
Идентификатор: ГЦК 087
Возраст: 21 год. Диагноз: ФлК.
Выполнен анализ наличия экспрессии DNAJB1-PRKACA заявленным способом.
Результат: При исследовании иссеченного тканевого материала пациента экспрессия DNAJB1-PRKACA обнаружена в ткани опухоли и не обнаружена в прилежащей условно здоровой ткани печени.
Пример 4.
Идентификатор: ГЦК 103
Возраст: 17 лет.
Диагноз: ФлК.
Выполнен анализ наличия экспрессии DNAJB1-PRKACA заявленным способом.
Результат: При исследовании иссеченного тканевого материала пациента экспрессия DNAJB1-PRKACA обнаружена в ткани опухоли и не обнаружена в прилежащей условно здоровой ткани печени.
Пример 5.
Идентификатор: ГЦК 108
Возраст 19 лет.
Диагноз: ФлК.
Выполнен анализ наличия экспрессии DNAJB1-PRKACA заявленным способом.
Результат: При исследовании иссеченного тканевого материала пациента экспрессия DNAJB1-PRKACA обнаружена в ткани опухоли, и не обнаружена в прилежащей условно здоровой ткани печени.
Пример 6.
Идентификатор: Г ЦК 144
Возраст: 30 лет.
Диагноз: ФлК.
Выполнен анализ наличия экспрессии DNAJB1-PRKACA заявленным способом.
Результат: При исследовании иссеченного тканевого материала пациента экспрессия DNAJB1-PRKACA обнаружена в ткани опухоли и не обнаружена в прилежащей условно здоровой ткани печени.
Пример 7.
Идентификатор: ГЦК 148
Возраст: 20 лет.
Диагноз: ФлК.
Выполнен анализ наличия экспрессии DNAJB1-PRKACA заявленным способом.
Результат: При исследовании иссеченного тканевого материала пациента экспрессия DNAJB1-PRKACA обнаружена в ткани опухоли и не обнаружена в прилежащей условно здоровой ткани печени.
--->
Перечень последовательностей олигонуклеотидов
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="Method for
detecting the DNAJB1-PRKACA chimeric transcript in tissue samples
from patients with fibrolamellar liver carcinoma by real-time
polymerase chain reaction.xml" softwareName="WIPO Sequence"
softwareVersion="2.2.0" productionDate="2023-03-09">
<ApplicationIdentification>
<IPOfficeCode> N.N. Blokhin National Medical Research Center of
Oncology оf the Ministry of Health of the Russian
Federation</IPOfficeCode>
<ApplicationNumberText> МПК:C12N15/00</ApplicationNumberText>
<ApplicantFileReference>МПК: C12N15/00</ApplicantFileReference>
<ApplicantName languageCode="ru">Наталия Лазаревич </ApplicantName>
<ApplicantNameLatin>Natalia Lazarevich</ApplicantNameLatin>
<InventorName languageCode="ru">Артем Горев</InventorName>
<InventorNameLatin>Artem Gorev</InventorNameLatin>
<InventionTitle languageCode="ru">Способ выявления химерного
транскрипта DNAJB1-PRKACA в образцах ткани пациентов с
фиброламеллярной карциномой печени методом полимеразной цепной
реакции в реальном времени</InventionTitle>
<SequenceTotalQuantity>6</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttcaaggagatcgctgaggc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgtgttctgagcgggacttt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgctacggggaggaagtgaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aaggatgcacccaacctcat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gatggtccttgttgacacagt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgagaggaggcggcacggga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ количественного определения мутаций F317L и F359V/C киназного домена BCR-ABL у больных хроническим миелоидным лейкозом, резистентных к терапии ингибиторами тирозинкиназ | 2019 |
|
RU2698461C1 |
| Способ количественного определения мутации G250E киназного домена BCR-ABL у больных хроническим миелоидным лейкозом, резистентных к терапии ингибиторами тирозинкиназ | 2016 |
|
RU2620076C1 |
| Способ выявления химерных транскриптов гена EWSR1, ассоциированных с саркомой Юинга | 2021 |
|
RU2759810C1 |
| Способ генотипирования полиморфного локуса rs4926222 (G>A) гена DNAJB1 у человека методом ПЦР в режиме "реального времени" с применением аллель-специфических флуоресцентных зондов | 2024 |
|
RU2829225C1 |
| СПОСОБ АНАЛИЗА ТРАНСЛОКАЦИЙ EML4-ALK, АССОЦИИРОВАННЫХ С ЧУВСТВИТЕЛЬНОСТЬЮ РАКА ЛЕГКОГО К ПРОТИВООПУХОЛЕВОЙ ТАРГЕТНОЙ ТЕРАПИИ | 2012 |
|
RU2509153C1 |
| Способ диагностики острых лейкозов и наборы для его выполнения | 2021 |
|
RU2806591C2 |
| СПОСОБ ИДЕНТИФИКАЦИИ ХРОМОСОМНЫХ ТРАНСЛОКАЦИЙ, ПРИВОДЯЩИХ К РАЗВИТИЮ ЗЛОКАЧЕСТВЕННЫХ ЗАБОЛЕВАНИЙ КРОВИ (ЛЕЙКОЗОВ), С ИСПОЛЬЗОВАНИЕМ ОЛИГОНУКЛЕОТИДНОГО БИОЛОГИЧЕСКОГО МИКРОЧИПА (БИОЧИПА) | 2004 |
|
RU2286798C2 |
| СПОСОБ ДИАГНОСТИКИ НЕМЕЛКОКЛЕТОЧНОГО РАКА ЛЕГКИХ И НАБОР ДЛЯ ЕГО ОСУЩЕСТВЛЕНИЯ | 2010 |
|
RU2445627C1 |
| ОПРЕДЕЛЕНИЕ СНИЖЕНИЯ УРОВНЯ мРНК ГЕНОВ ITGA9, HYAL1 И HYAL2 КАК СПОСОБ ДИАГНОСТИКИ НЕМЕЛКОКЛЕТОЧНОГО РАКА ЛЕГКОГО И НАБОР ДЛЯ ЕГО ОСУЩЕСТВЛЕНИЯ | 2008 |
|
RU2403575C2 |
| ОПРЕДЕЛЕНИЕ СНИЖЕНИЯ УРОВНЯ мРНК ГЕНА ZG16 КАК СПОСОБ ДИАГНОСТИКИ РАКА ТОЛСТОЙ КИШКИ И НАБОР ДЛЯ ЕГО ОСУЩЕСТВЛЕНИЯ | 2009 |
|
RU2395234C1 |
Изобретение относится к областям биотехнологии и медицины. Описан способ выявления химерного транскрипта DNAJB1-PRKACA в клинических образцах тканей пациентов с фиброламеллярной карциномой печени методом полимеразной цепной реакции в реальном времени, отличающийся использованием специфических олигонуклеотидных последовательностей - праймеров - SEQ ID NO: 1, NO: 2; SEQ ID NO: 4, NO: 5, дополненный специально адаптированными флуоресцентными зондами SEQ ID NO: 3, ID NO: 6. Изобретение позволяет дифференцировать фиброламеллярные карциномы от других типов опухолей печени для выбора оптимальной тактики лечения специфического заболевания, а также обеспечивает повышение точности диагностики за счет применения оригинальных олигонуклеотидных последовательностей и оптимизированных условий реакций. 2 ил., 7 пр.
Способ выявления химерного транскрипта DNAJB1-PRKACA в клинических образцах тканей пациентов с фиброламеллярной карциномой печени методом полимеразной цепной реакции в реальном времени, отличающийся использованием специфических олигонуклеотидных последовательностей - праймеров - SEQ ID NO: 1, NO: 2; SEQ ID NO: 4, NO: 5, дополненный специально адаптированными флуоресцентными зондами SEQ ID NO: 3, ID NO: 6.
| Д | |||
| А | |||
| Шавочкина, И | |||
| Ф | |||
| Кустова и др | |||
| Фиброламеллярная карцинома как отдельный подтип гепатоцеллюлярного рака: молекулярно-генетические особенности, диагностика, перспективы лечения; Д | |||
| А | |||
| Шавочкина, И | |||
| Ф | |||
| Кустова, Н | |||
| Л | |||
| Лазаревич | |||
| Фиброламеллярная карцинома как отдельный подтип гепатоцеллюлярного рака: молекулярно-генетические особенности, |
Авторы
Даты
2023-11-14—Публикация
2023-04-06—Подача