Изобретение относится к области молекулярной диагностики и может быть использовано для выявления транскриптов гена EWSR1 с целью диагностики саркомы Юинга.
Саркома Юинга (СЮ) представляет собой остеолитическое злокачественное новообразование, отличающееся крайне быстрым возникновением метастазов в легких, головном мозге, висцеральных органах. Преимущественно возникает у детей и подростков 10-15 лет, но также может встречаться у людей старшего и пожилого возраста [1]. Она составляет 10-15% всех первичных злокачественных неоплазий костной ткани у детей [2]. По данным ВОЗ, СЮ является наиболее распространенной детской опухолью костей и диагностируется с частотой 0,5 на 100000 населения ежегодно [3]. Риск возникновения заболевания повышается в период полового созревания, а также в период активного роста скелета. В 30% случаев опухоль локализуется в длинных трубчатых костях нижних конечностей, а именно в диафизе кости. К редким локализациям относится поражение ребер (6,5%), позвонков (5%), костей черепа (4%) и пястных костей (3,8%). Крайне редко поражается эпифиз трубчатых костей [3,4].
К семейству саркомы Юинга относятся также высокоагрессивные злокачественные новообразования, поражающие в основном костную ткань – примитивные нейроэктодермальные опухоли (PNETs), опухоль Аскина, внескелетная саркома Юинга [5]. Все опухоли данного семейства имеют схожий нейрогенный фенотип и возникают вследствие одной и той же хромосомной транслокации. Поэтому их рассматривают как варианты одной опухоли, имеющие разные степени дифференцировки [6].
В лечении рассматриваемых опухолей используются стандартные химиотерапевтические методы, которые являются весьма токсичными, вызывают много нежелательных побочных эффектов и далеко не всегда эффективны. Поэтому в настоящее время разрабатываются новые терапевтические подходы, к которым можно отнести ингибитор РНК-геликазы A (YK-4-279), который связывается с онкогенным белком EWSR1/FLI1, являющимся драйвером возникновения СЮ, что препятствует активации транскрипции, вызванной EWSR1/FLI [7]. Данный препарат сейчас находится на I фазе клинических исследований [NCT02657005].
Более 85% СЮ возникают вследствие транслокации t (11; 22) (q24; q12), в результате которой ген EWSR1, расположенный на длинном плече 22-й хромосомы, соединяется с геном FLI1, расположенным на длинном плече 11-й хромосомы. Более редко (10%-15% случаев) встречаются альтернативные транслокации, приводящие к слиянию гена EWSR1 с другими факторами транскрипции ETS: ERG, ETV1, ETV4, FEV и др. Всего описано около 20 различных типов транслокаций, характерных для СЮ и PNETs. Наиболее часто встречаются слияния генов EWSR1 и FLI1, EWSR1 и ATF1, EWSR1 и ERG, составляя около 75% от числа всех транслокаций. Более половины всех сарком Юинга имеют слияние типа I, при котором происходит соединение 5’-области мРНК EWSR1 экзона 7, с 3’-областью мРНК FLI1 экзона 5. Примерно в 25% случаев происходят слияния типа II, при котором экзон 6 FLI1 является партнером для экзона 7 EWSR1 [7].
Транслокация t (22; 11)(q; 24; 12) в дальнейшем приводит к экспрессии химерного онкопротеина EWSR1/FLI1, который уникально экспрессируется во всех опухолевых клетках и поддерживает их выживание [8,9]. Слияние EWSR1/FLI1 присутствует только в клетках СЮ и не существует ни в одной нормальной клетке организма. Таким образом, СЮ содержат уникальный белок, генерируемый опухолеспецифической транслокацией, который может служить мишенью для разработки таргетных препаратов, а описанная транслокация является специфичным диагностическим маркером [10].
В настоящее время для обнаружения транслокации доступны различные методы. Среди них флуоресцентная гибридизация in situ (FISH) является наиболее часто используемой в рутинной клинической диагностике. Однако FISH метод имеет ряд недостатков, к которым относится трудоемкость и моноплексность (выявление только одного транскрипта с помощью одной гибридизационной пробы). Для повышения информативности требуется проведение множественных гибридизаций, что существенно удлиняет время, отведенное на диагностику. Также отсутствие специального оборудования, необходимого для проведения данного метода в большинстве лабораторий, существенно снижает возможности диагностики. Кроме того, иногда бывает трудно выбрать конкретный зонд для FISH-теста, в случае неясного морфологического диагноза.
Поэтому разработка оптимального метода диагностики СЮ на сегодняшний день является актуальной задачей. Обнаружение одного из нескольких патогномоничных химерных транскриптов, лежащих в основе патогенеза сарком, является золотым стандартом диагностики. Это обуславливает обоснованность применения различных молекулярно-генетических методов для их выявления.
Молекулярные методы уже повсеместно включаются в рутинную диагностику.
Например, известен способ выявления транслокаций с помощью мультиплексной ПЦР и последующего высокопроизводительного секвенирования (NGS) (Archer Fusion Plex Sarcoma kit позволяет выявить перестройки сразу 26 генов). Обнаружение транслокаций с помощью целевого NGS на основе мультиплексной ПЦР показало удовлетворительные результаты по сравнению с традиционными FISH и RT-PCR на FFPE тканях при условии соблюдения критериев адекватного качества материала [11].
Известен способ выявления транскриптов EWSR1/ERG, EWSR1/FLI с помощью RT-PCR с раздельной стадией обратной транскрипции с детекцией результатов с помощью капиллярного электрофореза, при этом длина апликонов составляла около 200 п.н, что снижает чувствительность и эффективность RT-PCR. В качестве контроля выделения нуклеиновых кислот использовали амплификацию кДНК гена β-актина (ACTB) [12].
Известен способ выявления транскриптов EWSR/FLI1, EWSR1/ERG, и EWSR1/FEV с помощью RT-PCR с раздельной стадией обратной транскрипции с детекцией результатов с помощью электрофореза в 2%-номагарозном гель-электрофорезе. При этом длина ампликонов составляла около 250 п.н., что также снижает эффективность проводимой реакции амплификации. В качестве контроля выделения использовали амплификацию кДНК гена β-актина (ACTB) [13].
Известен способ выявления транскрипта EWSR1/FLI1 с помощью «вложенной» PCR с раздельной стадией обратной транскрипции с детекцией результатов с помощью электрофореза также на 2%-ном агарозном геле, что трудоемко и имеет низкую чувствительность [14].
Известен способ выявления транскриптов EWS/FLI1, EWS/ERG, EWS/ETV1 с помощью RT-PCR с раздельной стадией обратной транскрипции. Для детекции ампликонов использовали интеркалирующий краситель SYBR Green I, что может уменьшать специфичность детекции. При этом длина ампликонов была достаточно небольшой: от 80 до 166 п.н [15].
Патентов, описывающих выявление химерных транскриптов гена EWSR1 с помощью метода Real-time PCR, не было найдено.
Наиболее близким к заявляемому способу - прототипом, является способ выявления транскрипта EWSR1/FLI с помощью RT-PCR с раздельной стадией обратной транскрипции с детекцией результатов с помощью электрофореза в 2%-ном агарозном геле. Нуклеиновые кислоты выделяли при помощи набора RNeasy Mini Kit (Qiagen). При этом длина ампликонов составляла от 150 до 200 п.н. В качестве контроля выделения нуклеиновых кислот использовали амплификацию кДНК гена фосфоглицераткиназы (PGK) [16].
Недостатками известного способа являются проведение раздельной стадии обратной транскрипции, что значительно удлиняет время проведения анализа, а также выбранная длина ампликонов в районе 150-200 п.н., что существенно снижает эффективность амплификации.
Задачей изобретения является разработка менее трудоемкого и более быстрого способа выявления молекулярных перестроек гена EWSR1, обладающего высокой чувствительностью.
Технический результат: уменьшение длительности способа с сохранением высокой чувствительности диагностики, что является важным преимуществом для анализа нуклеиновых кислот, выделенных из парафиновых блоков.
Поставленная задача достигается путем проведения одностадийной Real-time PCR с детекцией продуктов амплификации специфическими флуоресцентными зондами (SEQ ID NO:3, SEQ ID NO:6, SEQ ID NO:9, SEQ ID NO:12, SEQ ID NO:15) для дифференциальной диагностики саркомы Юинга с максимальным сокращением времени анализа.
Предлагаемый способ заключается в следующем.
Для анализа используют образцы РНК, выделенные из фиксированной ткани саркомы Юинга в парафиновых блоках. Для выделения РНК используют свежеприготовленные срезы FFPE тканей толщиной 5 мкм, помещенные в пробирку объемом 1.5 мл. После депарафинизации тканей проводят выделение РНК методом фенол-хлороформной экстракции [17]. Образцы РНК, выделенные из фиксированной ткани саркомы Юинга в парафиновых блоках, подвергают одностадийной Real-time PCR с сопряженной реакцией обратной транскрипции. Для оценки успешности выделения РНК, стадий обратной транскрипции и амплификации используют амплификацию фрагмента кДНК гена Bcl-XL.
Уровень экспрессии генов в ходе проведения одностадийной Real-time PCR, сопряженной с реакцией обратной транскрипции, определяют на амплификаторе Light Cycler 96 (Roche, Швейцария) и Cfx 96 (Bio-Rad). Набор праймеров, используемых для детекции химерных транскриптов гена EWSR1, включает прямые праймеры с последовательностями SEQ ID NO:1, SEQ ID NO:4, SEQ ID NO:7, SEQ ID NO:10, SEQ ID NO:13 и обратные праймеры с последовательностями SEQ ID NO:2, SEQ ID NO:5, SEQ ID NO:8, SEQ ID NO:11, SEQ ID NO:14, а также зонды с последовательностями SEQ ID NO:3, SEQ ID NO:6, SEQ ID NO:9, SEQ ID NO:12, SEQ ID NO:15, представленные в перечне последовательностей.
Определяющими отличиями заявляемого способа, по сравнению с прототипом, являются:
1) стадию обратной транскрипции (ОТ) и полимеразную цепную реакцию в режиме реального времени проводят в одной реакционной смеси, что позволяет существенно уменьшить длительность и трудоемкость способа;
2) накопление продуктов амплификации детектируют при помощи оригинальных олигонуклеотидных проб длиной 20-23 п.н., на 5'-конце которых находится флуорофор (HEX), а на 3'-конце тушитель флуоресценции (BHQ-2), что обеспечивает максимальное уменьшение длины ампликона и приводит к увеличению эффективности амплификации, повышению чувствительности анализа;
3) используют наборы праймеров и зондов, включающие прямые праймеры с последовательностями SEQ ID NO:1, SEQ ID NO:4, SEQ ID NO:7, SEQ ID NO:10, SEQ ID NO:13, обратные праймеры с последовательностями SEQ ID NO:2, SEQ ID NO:5, SEQ ID NO:8, SEQ ID NO:11, SEQ ID NO:14, а также зонды с последовательностями SEQ ID NO:3, SEQ ID NO:6, SEQ ID NO:9, SEQ ID NO:12, SEQ ID NO:15, которые фланкируют область разрыва двух генов - fusion, расположенную между участками гена EWSR1 и гена ETS-семейства (FLI1, ATF1, ERG, NRA3, CREB1).
Изобретение иллюстрируется следующими примерами конкретного выполнения.
Пример 1. Выявление наиболее распространенного химерного транскрипта EWSR/FLI в FFPE блоках, содержащих ткань саркомы Юинга.
Для выделения РНК использовали свежеприготовленные срезы FFPE тканей толщиной 5 мкм, помещенные в пробирку объемом 1.5 мл. Для депарафинизации тканей в пробирку со срезами добавляли 500 мл минерального масла (Paraffin oil light), после чего инкубировали при 5 мин при 800С. Далее добавляли 200 мкл буфера А, 20 мкл proteinase К, перемешивали, скидывали капли на дно пробирки кратковременным центрифугированием на вортексе, инкубировали при 65оС 12 ч. После растворения осадка добавляли 200 мкл фенола, уравновешенного ТЕ, 200 мкл хлороформа, тщательно перемешивали до образования однородной эмульсии, инкубировали на льду в течение 10 мин. После этого центрифугировали в течение 10 мин при 13000 g. Далее 180 мкл верхней водной фазы переносили в чистые пробирки, содержащие 5 мкл линейного полиакриламида, 20 мкл 2М NaAc, перемешивали, добавляли 200 мкл изопропилового спирта. Полученный раствор инкубировали 1 час при температуре -20оС. Далее центрифугировали в течение 12 мин при 13 000 g. После этого удаляли супернатант, а оставшийся осадок дважды промывали 500 мкл буфера В. Далее сушили осадок в термостате при 37оС с открытыми крышками пробирок в течение 10 мин. Осадок растворяли в 80 мкл 5 mM Tris HCl pH 8,0 при 65оС в течение 20 мин. Немедленно использовали полученные препараты нуклеиновых кислот для анализа или хранили при -70оС.
Образцы РНК, выделенные из фиксированной ткани саркомы Юинга в парафиновых блоках, были подвергнуты одностадийной Real-time PCR, сопряженной с реакцией обратной транскрипции. Список обнаруженных перестроек приведен в таблице 1. Всего было проанализировано 30 образцов саркомы Юинга (СЮ).
Таблица 1
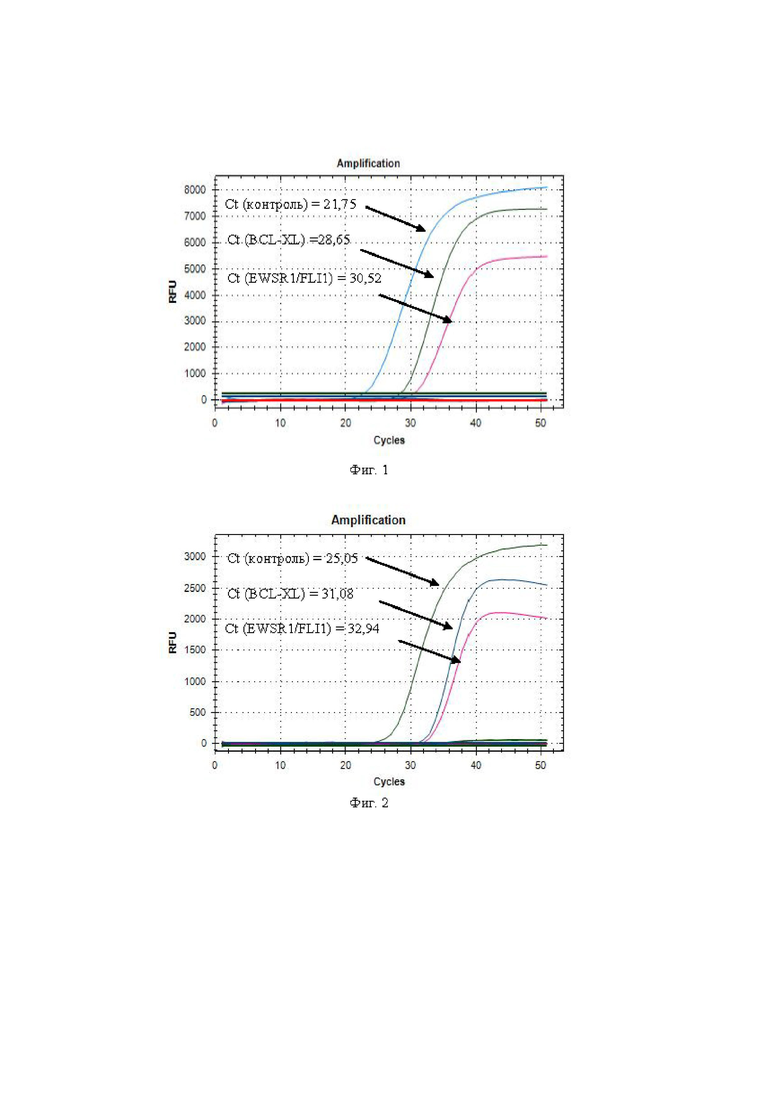
Для всех образцов была показана амплификация кДНК гена Bcl-XL, а также идентифицирована транслокация EWSR1/FLI по наличию амплификации со специфическими праймерами и зондами. Наглядный пример результатов проведения одностадийной Real-time PCR образца РНК, выделенного из FFPE ткани саркомы Юинга, представлен на фиг. 1, где кривые накопления флуоресцентного сигнала свидетельствуют о наличии перестройки EWSR1/FLI1 в образце СЮ.
Пример 2. Выявление химерных транскриптов гена EWSR1 и гена ETS-семейства (ATF1, ERG, NRA3, CREB1) в FFPE блоках, содержащих ткань саркомы Юинга
Образцы РНК были выделены из FFPE блоков аналогично примеру 1. Далее проводили одностадийную Real-time PCR с сопряженной реакцией обратной транскрипции. Для оценки успешности выделения РНК, стадий обратной транскрипции и амплификации использовали амплификацию фрагмента кДНК гена Bcl-XL. Список обнаруженных перестроек приведен в таблице 2. Всего было проанализировано 30 образцов саркомы Юинга (СЮ).
Таблица 2
Для всех образцов была показана амплификация кДНК гена Bcl-XL, а также идентифицированы транслокации EWSR1/ATF1, EWSR1/ERG, EWSR1/CREB1, EWSR1/NRA3 по наличию амплификации со специфическими праймерами и зондами. Наглядный пример результатов проведения одностадийной Real-time PCR образца РНК, выделенного из FFPE ткани саркомы Юинга, представлен на фиг. 2, где кривые накопления флуоресцентного сигнала свидетельствуют о наличии перестройки EWSR1/ATF1 в образце СЮ.
Использование предлагаемого способа позволит уменьшить время тестирования с сохранением высокой чувствительности диагностики, что является важным преимуществом для анализа нуклеиновых кислот, выделенных из парафиновых блоков.
Источники информации
1. Каприн А.Д., Распространенность злокачественных новообразований в России в 2007-2017 гг. // Состояние онкологической помощи населению России в 2017 году. Московский научно-исследовательский онкологический институт имени П.А. Герцена. - 2018. - 18 с.
2. Michaela C. Robust diagnosis of Ewing sarcoma by immunohistochemical detection of super-enhancer-driven EWSR1-ETS targets // Oncotarget. - 2017. - V. 9 - N 1. - P. 1587-1601.
3. Клинические рекомендации. Саркома Юинга у детей. (утв. Минздравом России). - 2017. - 15-18 с.
4. Кекеева Т.В. Анализ химерных онкогенов SYT/SSX1 и SYT/SSX2 при синовиальной саркоме // Молекулярная биология.- 2013. - Т. 45. - № 5.- С. 840-844.
5. Шалыга И.Ф., Ачинович С.Л. Саркома Юинга // Медицина и здравоохранение.- 2018. - Т.5.- № 54.- С. 102-105.
6. Miettinen M., Cupo W. Neural cell adhesion molecule distribution in soft tissue tumors // Human Pathology.–1993. – V. 24.– N 1. – P. 62-66.
7. Ingrid M. E. Desar. Systemic Treatment for Adults with Synovial Sarcoma // Curr Treat Options Oncol. - 2018. - V. 19 - N 2. - P. 22—28.
8. Catalog Of Somatic Mutations in Cancer, 2020
9. Denduluri S.K. Insulin-like growth factor (IGF) signaling in tumorigenesis and the development of cancer drug resistance. - 2015. - V. 2 - N 7. - P. 13-25.
10. Gaspar N. Ewing sarcoma: current management and future approaches through collaboration // J.Clin.Oncol. – 2015. – V. 33 – N 27. – P. 3036–3046.
Suk Wai Lam11. . Molecular Analysis of Gene Fusions in Bone and Soft Tissue Tumors by Anchored Multiplex PCR–Based Targeted Next-Generation Sequencing // The Journal of Molecular Diagnostics. Vol. 20,No 5, 2018, P. 653-663
12. Barbara Patócs. Utilisation of fluorescent multiplex PCR and laser induced capillary electrophoresis for the diagnosis of Ewing family of tumours in formalin-fixed paraffin-embedded tissues // J Clin Pathol. 2012; 65:1112–1118
13. Gamberi G. Molecular Diagnosis in Ewing Family Tumors// Molecular Diagnostics. - 2011. - V. 13 - N 3 - P.313–324.
14. Montoya C. Epigenetic control of the EWS/FLI1 promoter in Ewing's sarcoma//Oncology Reports 43: 1199-1207, 2020
15. Tracey B. L. Differentiating Ewing' sarcoma from other round blue cell tumors using a RT-PCR translocation panel on formalin-fixed paraffin-embedded tissues // Modern Pathology. - 2016. - V. 20 - N 5. - P. 397–404.
16. Berková A. A comparison of RT-PCR and FISH techniques in molecular diagnosis of Ewingʼs sarcoma in paraffin-embedded tissue/Čes.-slov. Patol. 44, 2008, No. 3, p. 67–70
17. Zumbo P. Phenol-chloroform Extraction. Weill Cornell Medical College.
--->
Перечень последовательностей
SEQ ID NO: 1
<110> Федеральное государственное бюджетное учреждение науки Институт
химической биологии и фундаментальной медицины Сибирского отделения
Российской академии наук (ИХБФМ СО РАН)
<120> Cпособ выявления химерных транскриптов гена EWSR1, ассоциированных
с саркомой Юинга.
<160> 15
<210> 1
<211> 19
<212> DNA
<213> Artificial sequence
<400> 1
CGTTTGTGCCCCTCCAAGG 19
SEQ ID NO: 2
<210> 2
<211> 20
<212> DNA
<213> Artificial sequence
<400> 2
CAACAGAGCAGCAGCTACGG 20
SEQ ID NO: 3
<210> 3
<211> 22
<212> DNA
<213> Artificial sequence
<400> 3
TCTGGCCTCAACAAAAGTCCTC 22
SEQ ID NO: 4
<210> 4
<211> 20
<212> DNA
<213> Artificial sequence
<400> 4
CTGTGCCTGGACTTGCCAAC 20
SEQ ID NO: 5
<210> 5
<211> 20
<212> DNA
<213> Artificial sequence
<400> 5
CAACAGAGCAGCAGCTACGG 20
SEQ ID NO: 6
<210> 6
<211> 22
<212> DNA
<213> Artificial sequence
<400> 6
CATTGCCCCAAATGGAGCCTTA 22
SEQ ID NO: 7
<210> 7
<211> 22
<212> DNA
<213> Artificial sequence
<400> 7
TCGGTACCATTGTTAGCCAGCT 22
SEQ ID NO: 8
<210> 8
<211> 20
<212> DNA
<213> Artificial sequence
<400> 8
CAACAGAGCAGCAGCTACGG 20
SEQ ID NO: 9
<210> 9
<211> 21
<212> DNA
<213> Artificial sequence
<400> 9
CATTACCCAGGGAGGAGCAAT 21
SEQ ID NO: 10
<210> 10
<211> 18
<212> DNA
<213> Artificial sequence
<400> 10
TGGGGTGGCCGTGACCGG 18
SEQ ID NO: 11
<210> 11
<211> 20
<212> DNA
<213> Artificial sequence
<400> 11
CAACAGAGCAGCAGCTACGG 20
SEQ ID NO: 12
<210> 12
<211> 22
<212> DNA
<213> Artificial sequence
<400> 12
TTACCATATGAGCCCCCCAGGA 22
SEQ ID NO: 13
<210> 13
<211> 22
<212> DNA
<213> Artificial sequence
<400> 13
GTGTGGGAGGTTGTATTATAGG 22
SEQ ID NO: 14
<210> 14
<211> 20
<212> DNA
<213> Artificial sequence
<400> 14
CAACAGAGCAGCAGCTACGG 20
SEQ ID NO: 15
<210> 15
<211> 21
<212> DNA
<213> Artificial sequence
<400> 15
CTCAGGGAAAGTTCACTGCTG 21
<---
Изобретение относится к биотехнологии, способ включает в себя совмещенную обратную транскрипцию образцов РНК, выделенных из фиксированной формальдегидом ткани в парафиновых блоках, и амплификацию их кДНК в режиме реального времени с использованием разработанного набора олигонуклеотидных праймеров и флуоресцентно меченных зондов. Изобретение позволяет проводить дифференциальную диагностику саркомы Юинга, обеспечивает уменьшение времени, затрачиваемого на проведение анализа, и снижение его стоимости. 1 з.п. ф-лы, 2 ил., 2 табл., 2 пр.
1. Способ выявления химерных транскриптов гена EWSR1, ассоциированных с саркомой Юинга, включающий проведение полимеразной цепной реакции в режиме реального времени с обратной транскрипцией (ОТ), отличающийся тем, что полимеразную цепную реакцию в режиме реального времени и стадию обратной транскрипции (ОТ) проводят в одной реакционной смеси, накопление продуктов амплификации детектируют в режиме реального времени при помощи набора праймеров и зондов, включающего прямые праймеры с последовательностями SEQ ID NO: 1, SEQ ID NO: 4, SEQ ID NO: 7, SEQ ID NO: 10, SEQ ID NO: 13, обратные праймеры с последовательностями SEQ ID NO: 2, SEQ ID NO: 5, SEQ ID NO: 8, SEQ ID NO: 11, SEQ ID NO: 14, а также зонды с последовательностями SEQ ID NO: 3, SEQ ID NO: 6, SEQ ID NO: 9, SEQ ID NO: 12, SEQ ID NO: 15.
2. Способ по п. 1, отличающийся тем, что в качестве матрицы для амплификации используют фрагмент кДНК гена Bcl-XL.
| Кекеева Т | |||
| В | |||
| и др, Химерные гены и транскрипты в онкологии, Молекулярная биология, 2011, Т | |||
| Железобетонный фасонный камень для кладки стен | 1920 |
|
SU45A1 |
| Кипятильник для воды | 1921 |
|
SU5A1 |
| Коммутатор для регулировочного автотрансформатора | 1921 |
|
SU793A1 |
| Wang W | |||
| L | |||
| et al., Detection and characterization of EWSR1/ATF1 and EWSR1/CREB1 chimeric transcripts in clear cell sarcoma (melanoma of soft parts), Modern Pathology, 2009, Т | |||
| Машина для добывания торфа и т.п. | 1922 |
|
SU22A1 |
| Разборный с внутренней печью кипятильник | 1922 |
|
SU9A1 |
| Электромагнитная вязальная машина | 1922 |
|
SU1201A1 |
| Rossi S | |||
| et al | |||
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
Авторы
Даты
2021-11-18—Публикация
2021-03-31—Подача