Область техники, к которой относится изобретение
Данное изобретение относится к биспецифическим антителам, содержащим антиген-связывающий домен, который связывается с рецептором трансферрина (TfR), и антиген-связывающий домен, который связывается с антигеном клеточной поверхности; к биспецифическим фрагментам антител; к ДНК, кодирующим указанные биспецифические антитела или биспецифические фрагменты антител; к векторам, включающим указанные ДНК; к гибридомам или трансформированным клеткам, продуцирующим указанные биспецифические антитела или биспецифические фрагменты антител; к способу получения указанных биспецифических антител или биспецифических фрагментов антител; к терапевтическим или диагностическим агентам, включающим указанные биспецифические антитела или биспецифические фрагменты антител; к способам лечения и диагностики, в которых используются указанные биспецифические антитела или биспецифические фрагменты антител, и к реагентам для выявления и количественного определения, включающие указанные биспецифические антитела или биспецифические фрагменты антител.
Предшествующий уровень техники
Антитела – это гликопротеины присутствующие в сыворотке или в тканевой жидкости у всех млекопитающих, и которые распознают чужеродные антигены в организме. Антитела участвуют в защите организма путем активации системы комплемента или эффекторных функций экспрессирующих FcR клеток, такие как фагоцитоз, зависимая от антител клеточно-опосредованная цитотоксичность (ADCC), высвобождение медиаторов и представление антигена посредством связывания с имеющимся на клеточной поверхности рецептором (FcR).
Молекула антитела состоит из четырех полипептидных цепей: двух гомологичных легких (L) и двух гомологичных тяжелых (H) и содержит два антиген-связывающих домена. Антитела подразделяют на классы и подклассы в зависимости от их тяжелых цепей; каждому классу и подклассу свойственны определенные функции. Известны пять различных классов антител человека: IgG, IgA, IgM, IgD и IgE. IgG дополнительно подразделяются на подклассы IgG1, IgG2, IgG3 и IgG4; а IgA дополнительно подразделяются на подклассы IgA1 и IgA2 (Charles A. J. et. al., Immunobiology, 1997, Current Biology Ltd/Garland Publishing, Inc.).
Поливалентным антителом называется антитело, в молекуле которого имеется несколько антиген-связывающих доменов. Примером поливалентного антитела является впервые описанное бивалентное антитело, которое моновалентно связывалось с каждым из двух различных антигенов, которое было получено в результате экспрессии в одной клетке H и L цепей, происходящих из двух различных типов антител, с помощью гибридомной технологии (NPL 1). Однако этим способом получается около десяти комбинаций H и L цепей антитела. Поэтому количество поливалентного антитела с желаемой комбинацией L и H цепей мало и, кроме того, трудно избирательно выделить и очистить нужное поливалентное антитело, так что выход желаемого антитела низкий.
Чтобы преодолеть эти трудности, были предприняты попытки получить антитело с желаемой комбинацией путем соединения нескольких антиген-связывающих доменов и экспрессии их как единой полипептидной цепи, чтобы сократить количество вариантов комбинирования субъединиц.
Например, известны антитела scFv, включающие единственную полипептидную цепь Fv, в которой антиген-связывающие домены L и H цепи объединены в один полипептид (NPL 2). Также сообщалось об антителах, в которых объединены два антиген-связывающих домена с использованием домена CH1 константной области тяжелой цепи IgG1 или частичного фрагмента этого домена, константной области легкой цепи или гибкого линкера (Gly-Gly-Gly-Gly-Ser), и о других подобных антителах (PTL1, PTL2).
Эти обычные поливалентные антитела имеют тот недостаток, что склонны к агрегации, а стабильность и продуктивность их получения невелики. Однако обнаружено, что те поливалентные антитела, которые включают несколько антиген-связывающих доменов в составе единого полипептида тяжелой цепи и в которых вариабельные области тяжелой цепи соединены друг с другом посредством линкера с аминокислотной последовательностью иммуноглобулинового домена или его фрагмента, обладают высокой стабильностью и их можно получать с высокой продуктивностью (PTL 3).
Рецептор трансферрина (в настоящем документе также обозначается TfR) был идентифицирован как компонент мембраны клеток, экспрессирующийся на поверхности ретикулоцитов. В функции TfR входит передача атомов железа, связанных с трансферрином (в настоящем документе обозначается Tf) внутрь клетки. Железо критически важно для поддержания клеточного гомеостаза и для пролиферации клеток, поэтому для поглощения железа рецептор трансферрина экспрессируется на высоком уровне в трофобластных клетках плаценты, в ретикулоцитах, в активированных лимфоцитах и в тех тканях, клетки которых в норме поглощают много железа. Известно также, что уровень экспрессии TfR высок в различных активно пролиферирующих опухолевых клетках (NPL 3 и 4). Также известно, что когда TfR перекрестно сшиваются на мембране клеток происходит интернализация образовавшегося комплекса путем эндоцитоза, зависимого от клатрина, и последующий распад TfR (NPL 5 и 6).
Так как уровень экспрессии TfR в нормальных клетках, за исключением клеток костного мозга, низкий, а в активно пролиферирующих раковых клетках высокий, рецептор трансферрина уже давно рассматривается как молекулярная мишень для лечения раковых заболеваний. Показано, что нацеленная на TfR молекула, например антитело, нейтрализующее рецептор трансферрина, весьма эффективна в качестве лекарства на моделях рака у мышей (PTL 4).
Известно, что в раковых клетках по сравнению с нормальными клетками усилена экспрессия определенных мембранных белков. Разработаны лекарственные средства, которые убивают только раковые клетки при помощи антител, избирательно распознающих такие мембранные белки. Например, в клинических испытаниях подтверждена эффективность лекарственных препаратов на основе антител, нацеленных на рецептор эпидермального фактора роста (далее называемого EGFR), при раке толстого кишечника или при раке головы и шеи (NPL 7).
EGFR – это рецептор типа тирозинкиназы, обнаруженный в качестве рецептора эпидермального фактора роста (далее называемого EGF). EGFR представляет собой трансмембранный гликопротеин имеющий молекулярную массу 170 кДа, при связывании своего лиганда образует димер и активирует последующий каскад внутриклеточного сигнального пути, тем самым способствуя пролиферации клеток, их дифференцировке, инвазии в ткани, метастазированию и проч.
EGFR экспрессируется в различных эпителиальных и мезенхимальных клетках и в клетках нервной ткани, но высокий уровень его экспрессии отмечается во многих раковых клетках, например в опухолях мозга и в плоскоклеточных карциномах (NPL 8 и 9); таким образом, EGFR считается опухолевым антигеном.
Установлено, что если использовать молекулу, связывающееся с двумя различными мембранными белками, то одна молекула антигена может служить стрежневой структурой а второй антиген может интернализоваться и подвергнуться деградации (PTL 5 и 6). Также сообщалось, что если пептид, распознающий специфический для раковых клеток мембранный белок, и пептид, распознающий TfR, химически связаны или объединены (слиты) при помощи метода гибридных белков, то в присутствии железо-хелатирующего агента, наблюдается подавление роста клеток, в которых экспрессируются оба этих антигена (PTL 7).
Список библиографических ссылок
Патентные публикации
PTL 1 - Заявка на патент США № 2007/0071675
PTL 2 - WO 2001/077342
PTL 3 - WO 2009/131239
PTL 4 - WO 2012/153707
PTL 5 - WO 2013/138400
PTL 6 - Заявка на Европейский патент № 2808035
PTL 7 - Патент США № 57025614
Не патентные публикации
NPL 1 - Suresh M.R. et. al., Methods Enzymol. 121, 210-228, 1986
NPL 2 – Kranz D.M. et. al., J. Hematother. 5, 403-408, 1995
NPL 3 - Hamilton T.A. et al., PNAS USA 76 6406-10, 1979
NPL 4 - Larson S.M. et al., J. Natl . Cancer Inst. 64 41-53, 1980
NPL 5 - Liu A.P. et al., J. Cell Biol. 191 1381-93, 2010
NPL 6 - Weissman A.M. et.al., J. Cell Biol. 102 951-8, 1986
NPL 7 – Инструкция по применению эрбитукса производства США (цетуксимаба) (в редакции от июня 2018 г.)
NPL 8 - Libermann T.A. et al., Cancer Res. 44 753-60, 1984
NPL 9 - Hendler F.J. et al., J. Clin. Invest. 74 647-51, 1984
Раскрытие изобретения
Техническая задача
Задача данного изобретения состоит в том, чтобы предложить новые биспецифические антитела, связывающися с TfR и с антигеном клеточной поверхности; биспецифические фрагменты антител; ДНК, кодирующие указанны биспецифические антитела или биспецифические фрагменты антител; векторы, включающие указанные ДНК; гибридомы или трансформированные клетки, продуцирующие указанные биспецифические антитела или биспецифические фрагменты антител; способ получения указанных биспецифических антител или биспецифических фрагментов антител; терапевтические или диагностические агенты, включающие указанные биспецифические антитела или биспецифические фрагменты антител; способы лечения и диагностики, в которых используются указанные биспецифические антитела или биспецифические фрагменты антител, и реагент для выявления или количественного определения, включающий указанные биспецифические антитела или биспецифические фрагменты антител.
Решение технической задачи
Для решения указанной выше задачи данным изобретением предлагается биспецифическое антитело или биспецифический фрагмент антитела, который связывается с TfR и с антигеном клеточной поверхности и в котором полипептид, содержащий домен, связывающийся с антигеном клеточной поверхности непосредственно (также называемый полипептидом N-концевой стороны), соединен непосредственно или через линкер с N-концевой стороной части молекулы IgG, которая связывается с TfR.
Итак, данное изобретение относится к следующему:
1. Биспецифическое антитело или биспецифический фрагмент антитела, которые связываются с TfR (TfR) и с антигеном клеточной поверхности и включают часть молекулы IgG, которая связывается с TfR, и полипептид N-концевой стороны, который связывается с антигеном клеточной поверхности, причем указанный полипептид N-концевой стороны непосредственно или через линкерную структуру связан с N-концом тяжелой цепи указанной части IgG.
2. Биспецифическое антитело или биспецифический фрагмент антитела по приведенному выше пункту 1, в котором часть IgG включает вариабельную область тяжелой цепи (VH), содержащую
- участок, определяющий комплементарность (CDR) 1, который включает аминокислотную последовательность, представленную SEQ ID NO: 32, или аминокислотную последовательность, являющуюся продуктом модификации последовательности SEQ ID NO: 32 путем по меньшей мере одной аминокислотной замены, выбираемой из замен тирозина в положении 2 на аланин или фенилаланин и треонина в положении 3 на аланин или глицин;
- участок CDR2, который включает аминокислотную последовательность, представленную SEQ ID NO: 33, или аминокислотную последовательность, являющуюся продуктом модификации последовательности SEQ ID NO: 33 путем по меньшей мере одной аминокислотной замены, выбираемой из замен валина в положении 1 на аланин, изолейцина в положении 2 на аланин или лейцин, валина в положении 7 на глутаминовую кислоту, аспарагиновой кислоты в положении 10 на аланин и аспарагиновой кислоты в положении 13 на пролин; и
участок CDR3, который включает аминокислотную последовательность, представленную SEQ ID NO: 34, или аминокислотную последовательность, являющуюся продуктом модификации последовательности SEQ ID NO: 34 путем по меньшей мере одной аминокислотной замены, выбираемой из замен глутамина в положении 3 на аланин или аспарагиновую кислоту, пролина в положении 4 на произвольно выбранную природную аминокислоту, триптофана в положении 5 на аланин, фенилаланин, гистидин или тирозин, тирозина в положении 7 на аланин или фенилаланин и валина в положении 13 на лейцин; и
вариабельную область легкой цепи (VL) имеющую CDR 1–3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 17 – 19 соответственно.
3. Биспецифическое антитело или биспецифический фрагмент антитела по приведенному выше пункту 1, в котором часть IgG включает одну вариабельную область тяжелой цепи (VH), выбираемую из приведенных ниже пунктов (ai) - (ci), и вариабельную область легкой цепи (VL) имеющую. CDR 1–3, содержащими аминокислотные последовательности, представленные SEQ ID NO: 17 – 19 соответственно;
(ai) VH содержащая CDR 1-3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 32-34 соответственно;
(bi) VH содержащая CDR 1-3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 42-44 соответственно;
(ci) VH содержащая CDR 1-3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 47 – 49 соответственно.
4. Биспецифическое антитело или биспецифический фрагмент антитела по приведенному выше пункту 1 или 2, в котором часть IgG включает VH, содержащую аминокислотную последовательность, представленную любой из последовательностей SEQ ID NO: 26, 31, 36, 41 и 46, и VL, содержащую аминокислотную последовательность, представленную SEQ ID NO: 16.
5. Биспецифическое антитело или биспецифический фрагмент антитела по приведенному выше пункту 1, в котором часть IgG включает VL содержащую CDR 1-3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 17-19 соответственно, и одну вариабельную область тяжелой цепи (VH), выбираемую из приведенных ниже пунктов (a) - (m):
(a) VH содержащая CDR 2 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 33 и 34 соответственно, и CDR1, который включает аминокислотную последовательность, являющуюся продуктом модификации последовательности SEQ ID NO: 32 путем замены тирозина в положении 2 на аланин или фенилаланин;
(b) VH содержащая CDR 2 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 33 и 34 соответственно, и CDR1, который включает аминокислотную последовательность, являющуюся продуктом модификации последовательности SEQ ID NO: 32 путем замены треонина в положении 3 на аланин или глицин;
(c) VH содержащая CDR 1 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 32 и 34 соответственно, и CDR2, который включает аминокислотную последовательность, являющуюся продуктом модификации последовательности SEQ ID NO: 33 путем замены валина в положении 1 на аланин;
(d) VH содержащая CDR 1 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 32 и 34 соответственно, и CDR2, который включает аминокислотную последовательность, являющуюся продуктом модификации последовательности SEQ ID NO: 33 путем замены изолейцина в положении 2 на аланин или лейцин;
(e) VH содержащая CDR 1 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 32 и 34 соответственно, и CDR2 который включает аминокислотную последовательность, являющуюся продуктом модификации последовательности SEQ ID NO: 33 путем замены валина в положении 7 на глутаминовую кислоту;
(f) VH содержащая CDR 1 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 32 и 34 соответственно, и CDR2, который включает аминокислотную последовательность, являющуюся продуктом модификации последовательности SEQ ID NO: 33 путем замены аспарагиновой кислоты в положении 10 на аланин;
(g) VH содержащая CDR 1 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 32 и 34 соответственно, и CDR2, который включает аминокислотную последовательность, являющуюся продуктом модификации последовательности SEQ ID NO: 33 путем замены аспарагиновой кислоты в положении 13 на пролин;
(h) вариабельная область тяжелой цепи с участками CDR 1 и 2, содержащие аминокислотные последовательности, представленные SEQ ID NO: 32 и 33 соответственно, и CDR3, который включает аминокислотную последовательность, являющуюся продуктом модификации последовательности SEQ ID NO: 34 путем замены глутамина в положении 3 на аланин или аспарагиновую кислоту;
(i) VH содержащая CDR 1 и 2, содержащие аминокислотные последовательности, представленные SEQ ID NO: 32 и 33 соответственно, и CDR3, который включает аминокислотную последовательность, являющуюся продуктом модификации последовательности SEQ ID NO: 34 путем замены пролина в положении 4 на произвольно выбранную природную аминокислоту;
(j) VH содержащая CDR 1 и 2, содержащие аминокислотные последовательности, представленные SEQ ID NO: 32 и 33 соответственно, и CDR3, который включает аминокислотную последовательность, являющуюся продуктом модификации последовательности SEQ ID NO: 34 путем замены пролина в положении 4 на аланин, тирозин, серин, аспарагиновую кислоту, глутамин, глутаминовую кислоту, треонин, аргинин, глицин, лизин, метионин, валин, лейцин, изолейцин, триптофан, фенилаланин или гистидин;
(k) VH содержащая CDR 1 и 2, содержащие аминокислотные последовательности, представленные SEQ ID NO: 32 и 33 соответственно, и CDR3, который включает аминокислотную последовательность, являющуюся продуктом модификации последовательности SEQ ID NO: 34 путем замены триптофана в положении 5 на аланин, фенилаланин, гистидин или тирозин;
(l) VH содержащая CDR 1 и 2, содержащие аминокислотные последовательности, представленные SEQ ID NO: 32 и 33 соответственно, и CDR3, который включает аминокислотную последовательность, являющуюся продуктом модификации последовательности SEQ ID NO: 34 путем замены тирозина в положении 7 на аланин или фенилаланин; и
(m) VH содержащая CDR 1 и 2, содержащие аминокислотные последовательности, представленные SEQ ID NO: 32 и 33 соответственно, и CDR3, который включает аминокислотную последовательность, являющуюся продуктом модификации последовательности SEQ ID NO: 34 путем замены валина на лейцин.
6 Биспецифическое антитело или биспецифический фрагмент антитела по приведенному выше пункту 1, в которых часть IgG включает VL, содержащую аминокислотную последовательность, представленную SEQ ID NO: 16, и VH, содержащую аминокислотную последовательность, являющуюся продуктом модификации аминокислотной последовательности. представленной SEQ ID NO: 31 путем по меньшей мере одной замены аминокислотного остатка, выбираемой из замен Y32A, Y32F, T33A, T33G, L45A, V48A, V50A, I51A, I51L, V55E, D58A, D61P, Q97A, Q97D, W99A, W99F, W99H, W99Y, Y100AA, Y100AF, V102L и замены P98 на произвольно выбранную аминокислоту (нумерация аминокислотных остатков указана в системе нумерации EU, согласованной с системой Кэбота (Kabat E.A. et al. 1991) далее в настоящем документе называется «индекс EU»).
7. Биспецифическое антитело или биспецифический фрагмент антитела по приведенному выше пункту 1, в которых часть IgG включает VL, содержащую аминокислотную последовательность, представленную SEQ ID NO: 16, и VH, содержащую аминокислотную последовательность, являющуюся продуктом модификации аминокислотной последовательности. представленной SEQ ID NO: 31 путем по меньшей мере одной замены аминокислотного остатка, выбираемой из замен Y32A, Y32F, T33A, T33G, L45A, V48A, V50A, I51A, I51L, V55E, D58A, D61P, Q97A, Q97D, W99A, W99F, W99H, W99Y, Y100AA, Y100AF, V102L, P98A, P98Y, P98S, P98D, P98Q, P98E, P98T, P98R, P98G, P98K, P98M, P98V, P98L, P98I, P98W, P98F и P98H (нумерация согласно индексу EU).
8. Биспецифическое антитело или биспецифический фрагмент антитела по приведенному выше пункту 1, в которых часть IgG распознает по меньшей мере один аминокислотный остаток, выбираемый из остатков Asp в положении 352, Ser в положении 355, Asp в положении 356 и Lys в положении 358, и по меньшей мере один аминокислотный остаток, выбираемый из остатков Met в положении 365, Val в положении 366 и Glu в положении 369 аминокислотной последовательности TfR, представленной SEQ ID NO: 6.
9. Биспецифическое антитело или биспецифический фрагмент антитела по любому из приведенных выше пунктов 1–8, в которых константная область тяжелой цепи части IgG содержит аминокислотную последовательность, представленную SEQ ID NO: 84 или SEQ ID NO: 86.
10. Биспецифическое антитело или биспецифический фрагмент антитела по любому из приведенных выше пунктов 1-9, в которых антигеном клеточной поверхности является EGFR или GPC.
11. Биспецифическое антитело или биспецифический фрагмент антитела по любому из приведенных выше пунктов 1-10, в которых антигеном клеточной поверхности является EGFR.
12. Биспецифическое антитело или биспецифическиспецифический фрагмент антитела по любому из приведенных выше пунктов 1-11, в которых полипептид N-концевой стороны представляет собой любой из фрагментов Fab, Fab’, scFv, dsFv и VHH антитела, направленного против данного антигена клеточной поверхности.
13. Биспецифическое антитело или биспецифический фрагмент антитела по любому из приведенных выше пунктов 1-12, в которых полипептид N-концевой стороны является фрагментом Fab антитела, направленного против данного антигена клеточной поверхности.
14. Биспецифическое антитело или биспецифический фрагмент антитела по любому из приведенных выше пунктов 1-9, в которых полипептид N-концевой стороны является фрагментом Fab антитела к EGFR, а антитело является антителом, включающим VH, содержащий CDR 1-3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 60-62 соответственно или SEQ ID NO: 65-67 соответственно, и VL, содержащий CDR 1-3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 17 -19 соответственно.
15. Биспецифическое антитело или биспецифический фрагмент антитела по приведенному выше пункту 14, в которых антитело к EGFR является антителом, выбираемым из описанного в пунктах (a) - (e):
(a) антитело, включающее VH, содержащую аминокислотную последовательность, представленную SEQ ID NO: 59 или SEQ ID NO: 64, и VL, содержащую аминокислотную последовательность, представленную SEQ ID NO: 16;
(b) антитело, включающее VH, содержащую аминокислотную последовательность, представленную SEQ ID NO: 110, и VL, содержащую аминокислотную последовательность, представленную SEQ ID NO: 111;
(c) антитело, включающее VH, содержащую аминокислотную последовательность, представленную SEQ ID NO: 112, и VL, содержащую аминокислотную последовательность, представленную SEQ ID NO: 113;
(d) антитело, включающее VH, содержащую аминокислотную последовательность, представленную SEQ ID NO: 114, и VL, содержащую аминокислотную последовательность, представленную SEQ ID NO: 115; и
(e) антитело, включающее VH, содержащую аминокислотную последовательность, представленную SEQ ID NO: 116, и VL, содержащую аминокислотную последовательность, представленную SEQ ID NO: 117.
16. Биспецифическое антитело или биспецифический фрагмент антитела по любому из приведенных выше пунктов 13-15, в которых С-конец тяжелой цепи фрагмента Fab непосредственно соединен с N-концом тяжелой цепи части IgG..
17. Биспецифическое антитело или биспецифический фрагмент антитела по любому из приведенных выше пунктов 13-15, в которых С-конец тяжелой цепи фрагмента Fab связан с N-концом тяжелой цепи части IgG посредством линкера.
18. Биспецифическое антитело или биспецифический фрагмент антитела по приведенному выше пункту 17, в котороых аминокислотная последовательность линкера состоит из всей аминокислотной последовательности шарнирного участка молекулы IgG или ее части..
19. Биспецифическое антитело или биспецифический фрагмент антитела по любому из приведенных выше пунктов 13-18, в которых аминокислотная последовательность, из которой состоит легкая цепь в фрагменте Fab, и аминокислотная последовательность, из которой состоит легкая цепь в части IgG, одинаковые.
20. ДНК, кодирующая биспецифическое антитело или биспецифический фрагмент антитела по любому из пунктов 1-19.
21. Рекомбинантный вектор, содержащий ДНК по приведенному выше пункту 20.
22. Трансформированная клетка, полученная путем введения рекомбинантного вектора по приведенному выше пункту 21 в клетку-хозяина.
23. Способ получения биспецифического антитела или биспецифического фрагмента антитела по любому из приведенных выше пунктов 1-19, включающий культивирование трансформированных клеток по пункту 22 в культуральной среде, обеспечивающей продуцирование этими клетками и накопление в их культуре биспецифического антитела или биспецифического фрагмента антитела по любому из пунктов 1-19, а также выделение указанного биспецифического антитела или биспецифического фрагмента антитела из этой культуры.
24. Терапевтический и/или диагностический агент, применяемый при заболеваниях, связанных по меньшей мере с одним из человеческого TfR или антигена клеточной поверхности, причем указанный агент включает в качестве активного ингредиента биспецифическое антитело или биспецифический фрагмент антитела по любому из пунктов 1-19.
25. Терапевтический и/или диагностический агент по приведенному выше пункту 24, в котором заболевание, связанное по меньшей мере с одним из человеческого TfR или антигена клеточной поверхности является раковым заболеванием.
26. Способ лечения и/или диагностики заболевания, связанного по меньшей мере с одним из человеческого TfR или антигена клеточной поверхности, в котором используется биспецифическое антитело или биспецифический фрагмент антитела по любому из приведенных выше пунктов 1-19.
27. Способ по приведенному выше пункту 26, в котором заболевание, по меньшей мере с одним из человеческого TfR или антигена клеточной поверхности, является раковым заболеванием.
28. Биспецифическое антитело или биспецифический фрагмент антитела по любому из приведенных выше пунктов 1-19 для применения при лечении или диагностике заболевания, связанного по меньшей мере с одним из человеческого TfR или антигена клеточной поверхности.
29. Биспецифическое антитело или биспецифический фрагмент антитела по приведенному выше пункту 28, в котором заболевание, связанное по меньшей мере с одним из человеческого TfR или антигена клеточной поверхности, является раковым заболеванием.
30. Применение биспецифического антитела или биспецифического антительного фрагмента по любому из приведенных выше пунктов 1-19 для получения терапевтического или диагностического агента, связанного по меньшей мере с одним из человеческого TfR или антигена клеточной поверхности.
31. Применение по приведенному выше пункту 30, в котором заболевание, связанное по меньшей мере с одним из человеческого TfR или антигена клеточной поверхности, является раковым заболеванием.
32. Реагент для выявления или количественного определения по меньшей мере одного из человеческого TfR или антигена клеточной поверхности, содержащий биспецифическое антитело или биспецифический фрагмент антитела по любому из приведенных выше пунктов 1-19
Полезные эффекты изобретения
По данному изобретению предлагаются новые биспецифические антитела и биспецифические фрагменты антител, связывающиеся с TfR и с антигеном клеточной поверхности; ДНК, кодирующие указанные биспецифические антитела и биспецифические фрагмент антител; векторы, содержащие указанные ДНК; гибридомы и трансформированные клетки, продуцирующие указанные биспецифические антитела и биспецифические фрагменты антител; способ получения указанных биспецифических антител и биспецифических фрагментов антител; терапевтические и диагностические агенты, включающие указанные биспецифические антитела и биспецифические фрагменты антител; способы лечения и диагностики с использованием указанных биспецифических антител и биспецифических фрагментов антител, и реагенты для выявления и количественного определения, содержащие указанные биспецифические антитела и биспецифические фрагменты антител.
Краткое описание чертежей
Фигура 1 изображает пример структуры биспецифического антитела по данному изобретению. На фиг. 1(А) показано биспецифическое антитело, в котором полипептидом N-концевой стороны является Fab а полипептид, включающий вариабельную область тяжелой цепи Fab, соединен с N-концом части IgG. На фиг. 1(В) показано биспецифическое антитело, в котором полипептидом N-концевой стороны является VHH. На фиг. 1(С) показано биспецифическое антитело, в котором в котором полипептидом N-концевой стороны является Fab и полипептид, включающий вариабельную область легкой цепи Fab, соединен с N-концом части IgG.
Фигура 2 представляет результаты определения уровня экспрессии определенного антигена в раковых клетках различных линий методом проточной цитометрии. На фиг. 2(А)-2(С) представлены результаты определения уровня экспрессии EGFR в клетках ОЕ21, T.Tn и U-937 соответственно, проточным цитометром. На фиг. 2(D)-2(F) представлены результаты определения уровня экспрессии TfR (TfR) в клетках ОЕ21, T.Tn и U-937, соответственно, проточным цитометром. По оси ординат – количество клеток, по оси абсцисс – интенсивность флуоресценции. Сплошная линия отражает аффинность связывания антитела к TfR или антитела к EGFR; гистограмма, закрашенная серым цветом, отражает аффинность связывания изотипического антитела, служившего отрицательным контролем.
Фигура 3 представляет результаты определения уровня экспрессии различных антигенов в раковых клетках печени методом проточной цитометрии. На фиг. 3(А)-3(С) показаны результаты определения уровня экспрессии GPC3 в клетках HepG2, HuH-7 и HLE, соответственно, пртоточным цитометром. На фиг. 3(D)-3(F) - результаты определения уровня экспрессии TfR в клетках HepG2, HuH-7 и HLE, соответственно, проточным цитометром. На фиг. 3(G)-3(I) - результаты определения уровня экспрессии EGFR в клетках HepG2, HuH-7 и HLE, соответственно, пртоточным цитометром. По оси ординат – количество клеток, по оси абсцисс – интенсивность флуоресценции. Сплошная линия отражает аффинность связывания антител к GPC3, или к TfR, или к EGFR; гистограмма, закрашенная серым цветом, отражает аффинность связывания изотипического антитела (или анти-DNP антитела, служивших отрицательным контролем.
Фигура 4 представляет результаты определения аффинности связывания различных антител (А)-(I) с клетками ОЕ21 методом проточной цитометрии. По оси ординат – количество клеток, по оси абсцисс – интенсивность флуоресценции. Сплошная линия отражает аффинность связывания антител к TfR или к EGFR; гистограмма, закрашенная серым цветом, отражает аффинность связывания вторичных антител, служивших отрицательным контролем.
Фигура 5 представляет результаты определения подавления роста клеткок ОЕ21 различными антителами к TfR. По оси ординат – жизнеспособность клеток, выраженная как отношение уровня ATP в живых клетках к его уровню в отрицательном контроле. Черные столбики – добавляли только моноклональные антитела, белые столбики – добавляли перекрестно-сшивающие антитела, соединяющие молекулы моноклонального антитела друг с другом.
Фигура 6 представляет результаты определения подавления роста клеток ОЕ21 каждым из антител к EGFR. По оси ординат – жизнеспособность клеток, выраженная как отношение уровня ATP в живых клетках к его уровню в отрицательном контроле.
Фигура 7A представляет результаты определения подавления роста клеток ОЕ21 роста различными биспецифическими антителами. По оси ординат – жизнеспособность клеток, выраженная как отношение уровня ATP в живых клетках к его уровню в отрицательном контроле.
Фигура 7B представляет результаты определения подавления роста клеток ОЕ21 роста различными биспецифическими антителами. По оси ординат – жизнеспособность клеток, выраженная как отношение уровня ATP в живых клетках к его уровню в отрицательном контроле.
Фигура 7C представляет результаты определения подавления клеток ОЕ21 различными биспецифическими антителами. По оси ординат – жизнеспособность клеток, выраженная как отношение уровня ATP в живых клетках к его уровню в отрицательном контроле.
Фигура 8A представляет результаты определения подавления роста линии раковых клеток ОЕ21, в которых экспрессируется EGFR, EGFR-TfR-биспецифическим антителом. По оси ординат – жизнеспособность клеток, выраженная как отношение уровня ATP в живых клетках к его уровню в отрицательном контроле.
Фигура 8B представляет результаты определения подавления роста линии раковых клеток T.Tn, в которых экспрессируется EGFR, EGFR-TfR-биспецифическим антителом. По оси ординат – жизнеспособность клеток, выраженная как отношение уровня ATP в живых клетках к его уровню в отрицательном контроле.
Фигура 8C представляет результаты определения подавления роста линии раковых клеток U-937, в которых экспрессируется EGFR, EGFR-TfR-биспецифическим антителом. По оси ординат – жизнеспособность клеток, выраженная как отношение уровня ATP в живых клетках к его уровню в отрицательном контроле.
Фигура 9 представляет результаты определения подавления роста клеток HepG2 GPC3-TfR-биспецифическими антителами. По оси ординат – жизнеспособность клеток, выраженная как отношение уровня ATP в живых клетках к его уровню в отрицательном контроле.
Фигура 10A представляет результаты определения подавления роста клеток HepG2, которыя евляются линией клеток рака печени в которых экспрессируется GPC3, GPC3-TfR-биспецифическим антителом. По оси ординат – жизнеспособность клеток, выраженная как отношение уровня ATP в живых клетках к его уровню в отрицательном контроле.
Фигура 10B представляет результаты определения подавления роста клеток HuH-7, которые являются линией клеток рака печени, в которых экспрессируется GPC3, GPC3-TfR-биспецифическим антителом. По оси ординат – жизнеспособность клеток, выраженная как отношение уровня ATP в живых клетках к его уровню в отрицательном контроле.
Фигура 10C представляет результаты определения подавления роста клеток HLE . которыя являются линией клеток рака печени, в которых экспрессируется GPC3, GPC3-TfR-биспецифическим антителом. По оси ординат – жизнеспособность клеток, выраженная как отношение уровня ATP в живых клетках к его уровню в отрицательном контроле.
Фигура 11 (A-E) представляет результаты определения ингибирования различными антителами или биспецифическими антителами связывания TfR с трансферрином на клетках OE21. По оси ординат – количество клеток, по оси абсцисс – интенсивность флуоресценции. Тонкая сплошная линия – активность связывания в условиях, когда флуоресцентно меченный трансферрин добавляли после добавления отрицательного контроля. Толстая сплошная линия – активность связывания в условиях, когда флуоресцентно меченный трансферрин добавляли после добавления антител или биспецифических антител. Гистограмма, закрашенная серым цветом, - контроль (без добавления флуоресцентно меченного трансферрина).
Фигура 12 представляет результаты определения эффекта на нисходящий сигнальный путь EGFR и на уровень экспрессии TfR при добавления EGFR-TfR-биспецифических антител к клеткам OE21 методом Вестерн-блоттинга. Каждая полоса отражает уровень экспрессии каждого белка, указанного в вертикальной строке справа при добавлении антител или биспецифических антител в концентрациях, указанных наверху. pEGFR – фосфорилированный EGFR; AKT –белок сигнального пути EGFR; pAKT - фосфорилированная AKT; TFRC – рецептор трансферрина; ACTB - β-актин.
Фигура 13. (A-D) представляет результаты определения методом проточной цитометрии влияния добавления EGFR-TfR-биспецифических антител к клеткам OE21 на уровень экспрессии TfR на клеточной поверхности По оси ординат – количество клеток, по оси абсцисс – интенсивность флуоресценции.. Тонкая сплошная линия – активность связывания в условиях, когда флуоресцентно меченный трансферрин добавляли после добавления отрицательного контроля и культивирования в течение 24 часов. Толстая сплошная линия – активность связывания в условиях, когда флуоресцентно меченный трансферрин добавляли после добавления антител или биспецифических антител и культивирования в течение 24 часов. Гистограмма, закрашенная серым цветом, - контроль (без добавления флуоресцентно меченного трансферрина).
Фигура 14 представляет результаты определение влияет ли истощение внутриклеточного железа на подавление роста клеток ОЕ21 EGFR-TfR-биспецифическими антителами. По оси ординат – жизнеспособность клеток, выраженная как отношение уровня ATP в живых клетках к его уровню в отрицательном контроле в условиях, когда добавляли PBS, и не добавляли FAS. Черные столбики - добавляли только антитело или биспецифическое антитело; заштрихованные столбики – вместе с антителом или биспецифическим антителом добавляли FAS.
Фигура 15 представляет результаты определения подавления роста клеток ОЕ21 при добавлении EGFR-TfR-биспецифических антител. По оси ординат – относительное содержание (%) клеток в лунке. Черные столбики – относительное содержание (%) клеткок в лунке при культивировании 7 суток в присутствии антитела или биспецифического антитела. Заштрихованные столбики - относительное содержание (%) клеткок в лунке при культивировании 7 суток в присутствии антитела или биспецифического антитела, и последующем удалении антитела посредством смены среды и культивировании еще 7 суток.
Фигура 16 представляет результаты определения подавления роста GEMM. при добавлении EGFR-TfR-биспецифических антител. По оси ординат – жизнеспособность клеток, где уровнь АТР в жизнеспособных клетках при добавлении PBS принимался за 100%.
Фигура 17A представляет результаты определения подавления клеточного роста различными EGFR-TfR-биспецифическими антителами.
Фигура 17B представляет результаты определения подавления клеточного роста различными EGFR-TfR-биспецифическими антителами.
Фигура 18 (А) схематически изображает структуру EGFR-TfR-биспецифических антител, полученных по Примеру 6, и (В) схематически изображает структуру гетеродимерных антител, полученных по Примеру 23.
Фигура 19A представляет результаты определения подавления клеточного роста различными EGFR-TfR-биспецифическими антителами.
Фигура 19B представляет результаты определения подавления клеточного роста различными EGFR-TfR-биспецифическими антителами.
Фигура 20 представляет результаты определения подавления клеточного роста различными EGFR-TfR-биспецифическими антителами, показателем чего служило подавление роста линии раковых клеток
Фигура 21 представляет результаты определения подавления клеточного роста различными EGFR-TfR-биспецифическими антителами.
Описание воплощений изобретения
Данное изобретение относится к биспецифическому антителу или биспецифическому фрагменту антитела, которые связываются с TfR и с антигеном клеточной поверхности, и в которых полипептид (называемый также полипептидом N-концевой стороны), содержащий антиген-связывающий домен, специфичный к антигену клеточной поверхности, соединен непосредственно или посредством линкера с N-концевой стороной части IgG, которая связывается с TfR (далее в настоящем документе эти антитела и фрагменты антител называются биспецифическими антителами и биспецифическими фрагментами антител по данному изобретению).
В настоящем документе обозначение TfR употребляется как синоним обозначений CD71, TFR1, TR, T9, p90 и IMD46. Примерами TfR являются человеческий TfR, содержащий аминокислотную последовательность, представленную в базе данных GenBank под регистрационным номером NP_003225 (http://www.ncbi.nlm.nih.gov/) или последовательностью SEQ ID NO: 6; обезьяний TfR, содержащий аминокислотную последовательность, представленную SEQ ID NO: 8, и подобные. Также к числу таких примеров относятся полипептиды, состоящие из аминокислотной последовательностей, являющейся продуктом делеции, замены или вставки одного или более аминокислотных остатков в аминокислотную последовательность, представленную SEQ ID NO: 6, последовательностью с регистрационным номером NP_003225 в базе данных GenBank или SEQ ID NO: 8, и обладающие функцией TfR .
Рецептор трансферрина по данному изобретению также включает полипептиды, содержащие аминокислотную последовательность, имеющие в целом 70% или больше, предпочтительно 80% или больше и более предпочтительно 90% или больше гомологию с аминокислотной последовательности, представленной SEQ ID NO: 6, последовательностью с регистрационным номером NP_003225 в базе данных GenBank или SEQ ID NO: 8, а наиболее предпочтительно – полипептиды, состоящие из аминокислотной последовательности, на 95%, 96%, 97%, 98%, 99% или более гомологичной указанной последовательности и обладающие функцией TfR .
Полипептид, аминокислотная последовательность которого является продуктом делеции, замены или вставки одного или более аминокислотных остатков в последовательности, представленной SEQ ID NO: 6, последовательности с регистрационным номером NP_003225 в базе данных GenBank или SEQ ID NO: 8, может быть получен, например, путем внесения сайт-специфических мутаций в ДНК, кодирующую SEQ ID NO: 6, последовательность с регистрационным номером NP_003225 в базе данных GenBank или SEQ ID NO: 8, методом сайт-специфического мутагенеза [Molecular Cloning, A Laboratory Manual, 2-е издание, Cold Spring Harbor Laboratory Press (1989), Current Protocols in Molecular Biology, John Wiley & Sons (1987-1997), Nucleic Acids Research, 10, 6487 (1982), Proc. Natl. Acad. Sci. USA, 79, 6409 (1982), Gene, 34, 315 (1985), Nucleic Acids Research, 13, 4431 (1985), Proceeding of the National Academy of Sciences in USA, 82, 488 (1985)] или подобным образом. Количество аминокислотных остатков, подлежащих делеции, замене или вставке, никак особо не ограничивается, но предпочтительно составляет от одного до нескольких десятков, например 1-20, более предпочтительно – от одного до десяти, например, 1-5.
Примерами гена, кодирующего TfR являются, например, нуклеотидная последовательность, кодирующая человеческоий TfR, представленная SEQ ID NO: 5 или последовательностью с регистрационным номером NM_003234 базе данных GenBank; нуклеотидная последовательность, кодирующая обезьяний TfR, представленная SEQ ID NO: 7, и другие подобные гены. Гены, кодирующие рецептор трансферрина по данному изобретению также включают гены, состоящие из нуклеотидной последовательности, являющейся продуктом делеции, замены или вставки одного или более нуклеотидов в нуклеотидной последовательности, представленной SEQ ID NO: 5, последовательности с регистрационным номером NM_003234 базе данных GenBank или SEQ ID NO: 7, и содержащие ДНК, кодирующую полипептид, обладающий функцией TfR ; гены, состоящие из нуклеотидной последовательности предпочтительно на 60% или больше, более предпочтительно на 80% или больше, еще более предпочтительно на 95% или больше гомологичной нуклеотидной последовательности, представленной SEQ ID NO: 5, последовательности с регистрационным номером NM_003234 базе данных GenBank или SEQ ID NO: 7, и содержащие ДНК, кодирующую полипептид, обладающий функцией TfR; гены, состоящие из ДНК, которая в жестких условиях гибридизуется с ДНК, состоящей из нуклеотидной последовательности, представленной SEQ ID NO: 5, последовательности с регистрационным номером NM_003234 в базе данных GenBank или SEQ ID NO: 7, и содержащие ДНК, кодирующую полипептид, обладающий функцией TfR.
Под ДНК, гибридизующейся в жестких условиях, в данном случае понимается, например, гибридизующаяся ДНК, получаемая при гибридизации колоний, гибридизации в бляшках, методом блоттинга по Саузерну или с ДНК-микрочипами, или другими методами, в которых в качестве зонда используют ДНК, имеющую нуклеотидную последовательность, представленную SEQ ID NO: 5 или последовательность с регистрационным номером NM_003234. Говоря конкретно, это, например, ДНК, которую идентифицируют путем промывания фильтра или предметного стекла при температуре 65°C раствором SSC в концентрации (0,1-2)х (состав раствора SSC в концентрации 1x: 150 ммоль/л хлорид натрия и 15 ммоль/л цитрат натрия) после гибридизации [Molecular Cloning, A Laboratory Manual, 2-е издание, Cold Spring Harbor Laboratory Press (1989), Current Protocols in Molecular Biology, John Wiley & Sons (1987-1997), DNA Cloning 1: Core Techniques, A Practical Approach, 2-е издание, Oxford University (1995)] при температуре 65°C в присутствии хлорида натрия в концентрации 0,7-1,0 моль/л с использованием фильтра или предметного стекла, на котором иммобилизована ДНК из используемых для гибридизации колоний или бляшек, или продукт полимеразной цепной реакции (ПЦР), или олигодезоксирибинуклеотид, имеющие указанную нуклеотидную последовательность. Примером способной к такой гибридизации ДНК является ДНК, предпочтительно на 60% или больше, более предпочтительно на 80% или больше, еще более предпочтительно на 95% или больше гомологичная нуклеотидной последовательности, представленной SEQ ID NO: 5 или последовательностью с регистрационным номером NM_003234.
У эукариот часто наблюдается полиморфизм нуклеотидных последовательностей генов, кодирующих белки. По данному изобретению в понятие «ген, кодирующий рецептор трансферрина» входят также полиморфные варианты гена, являющиеся продуктом небольших мутаций (замен, делеций или вставок одного или нескольких нуклеотидов) нуклеотидной последовательности.
Численное выражение гомологичности последовательностей по данному изобретению рассчитывается с помощью программ поиска гомологии, известных специалистам в данной области техники (если не оговорено иного); в частности, применительно к нуклеотидным последовательностям используются параметры по умолчанию программы BLAST [J. Mol. Biol., 215, 403 (1990)]; в случае аминокислотных последовательностей используются параметры по умолчанию программы BLAST 2 [Nucleic Acids Research, 25, 3389 (1997), Genome Research, 7, 649 (1997), http://www.ncbi.nlm.nih.gov/Education/BLASTinfo/information3.html] и подобные инструменты.
Что касается параметров по умолчанию, то штраф за открытие пропуска (G) в случае нуклеотидных последовательностей был 5, в случае аминокислотных последовательностей 11; штраф за продление пропуска (-E) в случае нуклеотидных последовательностей 2, в случае аминокислотных последовательностей 1; штраф за несовпадение нуклеотидов (-q) -3; бонус за совпадение нуклеотидов (-r) 1; ожидаемое число событий (-e) 10; длина слова (-W) в случае нуклеотидных последовательностей 11, в случае аминокислотных последовательностей 3; параметр (-y) [исключение (X) продлений в битах] в случае BLASTN 20, в случае иных программ семейства BLAST 7; параметр (-X ) (исключение X для выравнивания с пропусками в битах) 15, параметр -Z (конечное исключение X для выравнивания с пропусками в битах) в случае BLASTN 50, в случае иных программ семейства BLASТ 25 (http://www.ncbi.nlm.nih.gov/blast/html/blastcgihelp.html).
Полипептид, состоящий из части аминокислотной последовательности TfR, можно получить с помощью методов, известных специалистам в данной области техники; например, вырезают часть ДНК, кодирующую аминокислотную последовательность, соотвествующую SEQ ID NO: 6, последовательность с регистрационным номером NP_003225 в базе данных GenBank или SEQ ID NO: 8, и культивируют клетки, трансформированные вектором, содержащим полученную ДНК. Также можно получить, например, полипептид с аминокислотной последовательностью, являющейся продуктом делеции, замены или вставки одного или более аминокислотных остатков в части аминокислотной последовательности, представленной SEQ ID NO: 6, последовательности с регистрационным номером NP_003225 в базе данных GenBank или SEQ ID NO: 8; для этого применяют тот же способ, который описан выше, исходя из полипептида или ДНК, полученных описанным выше способом. Кроме того, полипептид, состоящий из части аминокислотной последовательности TfR, или полипептид с аминокислотной последовательностью, являющейся продуктом делеции, замены или вставки одного или более аминокислотных остатков в части аминокислотной последовательности TfR, можно получить путем химического синтеза, применяя, например, метод с использованием фторенилметилоксикарбонила (Fmoc) или трет-бутилоксикарбонила (tBoc).
С помощью программ, позволяющих предсказать трансмембранные области белка, а именно SOSUI (http://sosui.proteome.bio.tuat.ac.jp/sosuiframe0.html), TMHMM (версия 2) (http://www.cbs.dtu.dk/services/TMHMM-2.0/), ExPASy Proteomics Server (http://Ca.expasy.org/) и других подобных инструментов, можно определить внеклеточный домена TfR по данному изобретению, например, определить соотвествующую область в аминокислотной последовательности человеческого TfR, представленной последовательностью с регистрационным номером NP_003225 в базе данных GenBank Говоря конкретно, это часть аминокислотной последовательности, представленной SEQ ID NO: 2, или последовательности с регистрационным номером NP_003225 в базе данных GenBank, с 89-го положения по 760-е.
Примером функции TfR является поглощение железа, жизненно важное для жизнедеятельности и пролиферации клеток. Когда комплекс железа и трансферрина связывается с TfR, он поступает внутрь клетки путем эндоцитоза. В эндосоме рН понижается, и железо высвобождается из комплекса с трансферрином и переходит в цитоплазму при участии белка - транспортера двухвалентных ионов металлов (DMT1), где используется при пролиферации клетки или образовании энергии. Известно, что комплекс TfR и его рецептора, откуда высвобождается железо, как правило после этого не распадается и возвращается на клеточную поверхность (Yamashiro D. J. et al., Cell, 37 789-800, 1984).
Примеры клеток, в которых экспрессируется рецептор трансферрина, включают клетки многих нормальных тканей, в том числе костного мозга и плаценты; раковые клетки многих различных типов, в том числе опухолей толстого кишечника, головы и шеи, головного мозга, кроветворной ткани, печени и пищевода, а такжде многих линий раковых клеток, например HT29, HSC-2, RAMOS, K562, HepG2, OE21, T.Tn, U-937, HuH-7 и HLE.
Термин «антиген клеточной поверхности» по данному изобретению относится к антигенам, отличным от TfR, из числа антигенов, таких как белки и полипептиды, экспрессирующиеся на клеточной мембране раковых клеток или подобные им. Антиген клеточной поверхности по данному изобретению может быть любым при условии, что белок или полипептид, примером которых является антитело, может с ним связаться, но предпочтительно это такой антиген клеточной поверхности, который интернализуется и/или подвергается распаду путем связывания с белком или полипептидом, например с антителом. Также антиген клеточной поверхности по данному изобретению предпочтительно является белком с высоким уровнем экспрессии в раковых клетках или в тех популяциях клеток, которые участвуют в том или ином заболевании. Предпочтительные примеры антигенов клеточной поверхности по данному изобретению включают EGFR, GPC3 и т.п.
Обозначение EGFR в настоящем документе употребляется как синоним обозначений ERBB, ERBB1, HER1, PIG61, MENA, NISBD2, SA7 и c-Erb-1.
Примером EGFR является, например, человеческий EGFR, содержащий аминокислотную последовательность, представленную последовательностью с регистрационным номером NP005219 в GenBank или SEQ ID NO: 14; обезьяний EGFR, содержащий аминокислотную последовательность, представленную последовательностью с регистрационным номером XP_005549616.1 в GenBank. К числу примеров ЕGFR принадлежат также полипептиды с аминокислотной последовательностью, являющейся продуктом делеции, замены или вставки одногол или более аминокислотных остатков в аминокислотной последовательности, представленной SEQ ID NO: 14 или последовательности с регистрационными номерами NP005219 и XP_005549616.1 в GenBank и обладающие функцией EGFR.
EGFR по данному изобретению включает также полипептиды, имеющие предпочтительно 70% или больше, более предпочтительно 80% или больше, еще более предпочтительно 90% или больше гомологии с аминокислотной последовательностью, представленной SEQ ID NO: 14 или последовательностями с регистрационными номерами NP005219 и XP_005549616.1 в GenBank, и наиболее предпочтительно включает полипептиды, состоящие из аминокислотной последовательности, имеющей 95%, 96%, 97%, 98% и 99% или больше гомологии с указанной аминокислотной последовательности и обладающие функцией EGFR.
Полипептид, аминокислотная последовательность которого является продуктом делеции, замены или вставки одного или более аминокислотных остатков в последовательности, представленной SEQ ID NO: 14 или последовательностями с регистрационными номерами NP005219 и XP_005549616.1 в GenBank, может быть получен, например, путем внесения сайт-специфических мутаций в ДНК, кодирующую аминокислотную последовательность, представленную SEQ ID NO: 14 или последовательностями с регистрационными номерами NP005219 и XP_005549616.1 в GenBank, методом сайт-специфического мутагенеза или подобным образом. Количество аминокислотных остатков, подлежащих делеции, замене или вставке, никак особо не ограничивается, но предпочтительно составляет от одного до нескольких десятков, например 1-20, более предпочтительно – от одного до нескольких, например, 1-5.
Примером гена, кодирующего EGFR по данному изобретению, является, например, ген человеческого EGFR, содержащий нуклеотидную последовательность представленную SEQ ID NO: 13 или последовательностью, зарегистрированной в GenBank под номером NM_005228.
Ген, кодирующий EGFR, по данному изобретению включает также гены, состоящие из нуклеотидной последовательности, являющейся продуктом делеции, замены или вставки одного или более нуклеотидов в последовательности, представленной SEQ ID NO: 13 или последовательности, зарегистрированной в GenBank под номером NM_005228, и содержащие ДНК, которая кодирует полипептид, имеющий функцию EGFR; гены, состоящие из нуклеотидной последовательности, на 60% или больше, предпочтительно на 80% или больше, более предпочтительно на 95% или больше гомологичной нуклеотидной последовательности, представленной SEQ ID NO: 13 или последовательностью, зарегистрированной в GenBank под номером NM_005228, и содержащие ДНК, кодирующую полипептид, обладающий функцией EGFR; гены состоящие из ДНК, которая в жестких условиях гибридизуется с ДНК, содержащей нуклеотидную последовательность, представленную SEQ ID NO: 13 или последовательность, зарегистрированную в GenBank под номером NM_005228, и которая содержит ДНК, кодирующую полипептид, обладающий функцией EGFR, и подобные гены.
С помощью программ, позволяющих предсказать трансмембранные области белка, а именно SOSUI (http://sosui.proteome.bio.tuat.ac.jp/sosuiframe0.html), TMHMM (версия 2) (http://www.cbs.dtu.dk/services/TMHMM-2.0/), ExPASy Proteomics Server (http://Ca.expasy.org/) и других подобных инструментов, установлен внеклеточный домена EGFR по данному изобретению, например соотвествующая область в аминокислотной последовательности человеческого EGFR, представленной последовательностью, зарегистрированной в базе данных GenBank под номером NP005219. Говоря конкретно, это часть аминокислотной последовательности, представленной SEQ ID NO: 10 или последовательностьи, зарегистрированной в базе данных GenBank под номером NP005219, с 25-го положения по 645-е.
Функция EGFR состоит в связывании EGF, после чего образуется димер, и в результате запускаются сигнальные пути Ras/Raf/MAPK, PI3K/Akt или Jak/STAT, способствующие пролиферации кдеток, выживанию клеток, миграции клеток и их инвазии. Известно, что после связывания с эпидермальным фактором роста его рецептор интернализируется и, попав в результате эндоцитоза в лизосомы, расщепляется там (Ebner R et al., Cell Regul. 2 599-612, 1991).
Примеры клеток, в которых экспрессируется EGFR, включают эпителиальные клетки нормальных тканей, в том числе кожи, толстого кишечника и легких, а также эпителиальные раковые клетки, в том числе опухолей толстого кишечника, головы и шеи, легких и пищевода, и клетки раковых клеточных линий, например HT29, HSC-2, NCI-H1975, OE21, T.Tn, и A431.
В настоящем документе обозначение GPC3 употребляется как синоним обозначений DGSX, GTR2-2, MXR7, OCI-5, SDYS, SGB, SGBS и SGBS1.
В функции GPC3 входит связывание с белком, ассоциированным с сигнальным путем Wnt или Frizzled, и в результате он вовлечен в клеточное деление или пролиферацию раковых клеток, например рака печени.
Примером GPC3 является человеческий GPC3, содержащий аминокислотную последовательность, зарегистрированную в базе данных GenBank под номером P51654.
С помощью программ, позволяющих предсказать трансмембранную область белка, а именно SOSUI (http://sosui.proteome.bio.tuat.ac.jp/sosuiframe0.html), TMHMM (версия 2) (http://www.cbs.dtu.dk/services/TMHMM-2.0/), ExPASy Proteomics Server (http://Ca.expasy.org/) и других подобных инструментов, установлен внеклеточный домена GPC3 по данному изобретению, например соотвествующая область в аминокислотной последовательности человеческого GPC3, представленной последовательностью, зарегистрированной в базе данных GenBank под номером P51654. Говоря конкретно, это аминокислотная последовательность, представленная последовательностью, зарегистрированной в базе данных GenBank под номером P51654, с 1-го положения по 559-е.
Примеры клеток, в которых экспрессируется GPC3, включают эмбриональные клетки печени, а также раковые клетки, например клетки рака печени, и линий клеток рака печени, например HepG2 и HuH-7
Антитело является белком «произошедшим из» гена (обозначаемого как ген антитела), кодирующего целиком или частично вариабельную область тяжелой цепи, константную область тяжелой цепи, вариабельную область легкой цепи и константную область легкой цепи, которые образуют молекулу иммуноглобулина. По данному изобретению понятие антитело включает антитела или фрагменты антител любого класса или подкласса иммуноглобулинов.
Термин «тяжелая цепь» (обозначается H цепь) относится к полипептиду с более высокой молекулярной массой по сравнению с полипептидом другого типа из двух, образующих молекулу иммуноглобулина. Тяжелая цепь определяет класс и подкласс антитела. Выделяют пять классов антител: IgA, IgD, IgE, IgG и IgM соответственно пяти типам тяжелых цепей α, δ, ε, γ и μ, различающихся по аминокислотной последовательности константной области. Термин «легкая цепь» (обозначается L цепь) относится к полипептиду с меньшей молекулярной массой по сравнению с полипептидом другого типа из двух, образующих молекулу иммуноглобулина. У человека существуют два типа легких цепей - κ и λ.
Вариабельной (V) областью обычно называют область с высокой вариабельностью на стороне N-конца молекулы иммуноглобулина,. Поскольку структура остальной части варьирует меньше, ее называется константной (C). Соответствующие вариабельные области тяжелой и легкой цепей ассоциируются с формированием антиген-связывающего домена, определяющего способность антитела связываться с антигеном.
В тяжелой цепи человеческих антител вариабельной является часть аминокислотной последовательности с 1-го положения по 117-е (нумерация аминокислотных остатков указана в системе нумерации EU, согласованной с системой Кэбота (Kabat et al., Sequences of proteins of immunological interest, 199; 5-е издание), a константная область - это участок полипептидной цепи ниже 118-го положения. В легкой цепи человеческих антител вариабельным является участок с 1-го по 107-е положения аминокислотной последовательности (нумерация аминокислотных остатков по Кэботу), a константная область - это участок полипептидной цепи ниже 108-го положения. Вариабельные области тяжелой и легкой цепей обозначаются VH или VL соответственно
Антиген-связывающий домен – это домен, распознающий антиген и связывающийся с ним; этот термин относится к доменам, образующем конформацию, которая комплементарна антигенной детерминанте (эпитопу). Антиген-связывающий домен обеспечивают сильное межмолекулярное взаимодействие с антигенной детерминантой. Антиген-связывающий домен молекулы антитела образован вариабельными областями легких и тяжелых цепей; имеющих по меньшей мере три гипервариабельных участка, определяющих комплементарность с эпитопом (участки CDR). В человеческих антителах каждая из VH и VL имеет по три участка CDR, которые обозначаются CDR1, CDR2 и CDR3, считая от N-конца полипептидной цепи..
В константной области; константная область тяжелой и легкой цепи обозначаются CH и CL соответственно. Константные области тяжелых цепей классифицируют как α цепь, δ цепь ε цепь , γ цепь, и μ цепь. Константная область тяжелой цепи состоит из доменов СН1, шарнирного домена, CH2 и CH3 (указаны от N-конца полипептидной цепи), причем домены CH2 и CH3 вместе образуют область, называемую Fc. В свою очередь константные области легких цепей классифицируют на Cλ цепь и и Cκ цепь.
Моноклональные антитела – это антитела, секретируемые продуцирующими антитела клетками, поддерживающими моноклональность, и распознающие один определенный эпитоп (антигенную детерминанту). У всех молекул моноклонального антитела одинаковая аминокислотная последовательность (первичная структура) и одинаковая пространственная структура. Поликлональные антитела – это популяция молекул антител, секретируемых продуцирующими антитела клетками разных клонов. Олигоклональными антителами называют популяцию молекул антител, в которой смешаны различные моноклональные антитела.
Эпитоп – это структурный домен антигенной молекулы, который распознается антителом и связывается им. Примеры эпитопов включают отдельную аминокислотную последовательность, пространственную структуру, образованную аминокислотной последовательностью; аминокислотную последовательность, к которой присоединена цепь на основе сахаров; структуру, образованную аминокислотной последовательностью, к которой присоединена сахарная цепь, и тому подобное, каждая из которых распознается и связывается моноклональным антителом.
Примеры моноклональных антител по данному изобретению включают антитела, продуцируемые гибридомами, и генно-инженерные рекомбинантные антитела, продуцируемые клетками, которые трансформированы экспрессионным вектором, включающим ген антитела.
Гибридома может быть получена, например, путем получения антигена, получения клетки, продуцирующей антитело, обладающее антигенной специфичностью, из животного, иммунизированного антигеном, и затем слияния продуцирующей антитело клетки с клеткой миеломы. Желаемое моноклональное антитело может быть получено путем культивирования гибридомы или введения гибридомы животному для преобразования гибридомы в асцитную опухоль, отделения культурального раствора или асцита с последующей очисткой. В качестве животного для иммунизации антигеном, можно использовать любое животное, если оно может продуцировать гибридому, однако предпочтительно использовать мышь, крысу, хомяка, кролика или тому подобное. Кроме того, гибридома также может быть получена путем получения клетки, обладающей способностью продуцировать антитела, от такого иммунизированного животного, иммунизации этой клетки in vitro и последующего слияния этой клетки с клеткой миеломы.
Примеры генно-инженерного рекомбинантного антитела по данному изобретению включают антитела, полученные с использованием технологии рекомбинантных ДНК, например рекомбинантные мышиные антитела, рекомбинантные крысиные антитела, рекомбинантные антитела хомяка, рекомбинантные кроличьи антитела, человеческие химерные антитела (в настоящем документе называемые также просто химерными антителами), гуманизированные антитела (называемые также антителами с привитыми CDR) и человеческие антитела. В зависимости от вида животных, которые будут использоваться в качестве мишени и цели, для генно-инженерного рекомбинантного антитела можно определить применимые вариабельные области тяжелой цепи и легкой цепи и константные области, полученные из данного вида животного. Например, когда вид животного, который будет использоваться в качестве мишени, представляет собой человека, в качестве вариабельной области может быть принята область, полученная от человека или животного, не являющегося человеком, такого как мышь, и в качестве константной области и линкера могут быть приняты, те, что получены от человека.
Химерное антитело относится к антителу, состоящему из VH и VL антитела животного, отличного от человека (животное, не являющегося человеком), и CH и CL человеческого антитела. В качестве животного, не являющегося человеком, можно использовать любое животное, такое как мышь, крыса, хомяк или кролик, если оно может продуцировать гибридому. Химерное антитело можно получить путем получения кДНК, кодирующих VH и VL, из гибридомы, полученной от животного, не являющегося человеком, которое продуцирует моноклональное антитело, путем вставки каждой из кДНК в вектор экспрессии для животной клетки, имеющей ДНК, кодирующие CH и CL человеческого антитела, тем самым конструируя вектор экспрессии химерного антитела, а затем вводя вектор в клетку животного, чтобы вызвать экспрессию.
Гуманизированное антитело - это антитело, в котором CDR из VH и VL антитела не относящегося к человеку животного привиты в место соответствующих CDR из VH и VL человеческого антитела. Область, отличная от CDR из VH и VL, называется каркасной областью (в дальнейшем именуемой FR). Гуманизированное антитело можно получить путем конструирования кДНК, кодирующей аминокислотную последовательность VH, состоящую из аминокислотных последовательностей CDR из VH антитела животного, отличного от человека, и FR из VH произвольного человеческого антитела, и кДНК, кодирующих аминокислотную последовательность VL, состоящую из аминокислотной последовательности CDR из VL антитела животного, отличного от человека и аминокислотной последовательности FR из VL произвольного человеческого антитела, вставляя каждую из кДНК в вектор экспрессии в животной клетке, содержащий ДНК, кодирующие CH и CL человеческого антитела, тем самым конструируя вектор экспрессии гуманизированного антитела, а затем вводя вектор в животную клетку, чтобы вызвать экспрессию.
Термин «человеческое антитело» изначально относится к антителам, от природы присутствующим в организме человека, но он также включает антитела, полученные из фаговых библиотек человеческих антител и из трансгенных животных, продуцирующих человеческие антитела, получение которых стало возможным благодаря последним достижениям генной, клеточной или тканевой инженерии и тому подобного.
Чтобы получить антитела, от природы присутствующие в организме человека, можно, например, заразить лимфоциты периферической крови вирусом EB или подобным, для иммортализации лимфоцитов, и клонировать эти лимфоциты, чтобы получить культуру лимфоцитов, продуцирующих нужные антитела, которые выделяют из культурального супернатанта и очищают.
Фаговая библиотека человеческих антител представляет собой библиотеку, в которой фрагмент антитела, такой как фрагменты Fab или scFv, экспрессируется на поверхности фага за счет вставки гена антитела, полученного из В-клетки человека, в ген фага. Можно выделить фаг, который экспрессирует на поверхности фрагмент антитела, имеющий желаемую антигенсвязывающую активность, из библиотеки, используя активность связывания с субстратом, на котором иммобилизован антиген, в качестве показателя. Фрагмент антитела может быть дополнительно преобразован в молекулу человеческого антитела, состоящую из двух полных H-цепей и двух полных L-цепей, с использованием метода генной инженерии.
Трансгенное животное, продуцирующее человеческие антитела, - это животное, у которого ген человеческого антитела включен в клетку. В частности, например, трансгенная мышь, продуцирующая человеческие антитела, может быть получена путем введения гена человеческого антитела в мышиные клетки ES, имплантации этой ES-клетки в эмбрион мыши на ранних сроках с последующим выращиванием из эмбриона индивидуума. Человеческое антитело, полученное из трансгенного животного, продуцирующего человеческие антитела, может быть получено путем получения гибридомы с использованием обычного способа получения гибридомы, который выполняется с использованием животного, не являющегося человеком, и культивирования гибридомы, тем самым продуцируя и накапливая антитело в культуральном супернатанте.
CH генно-инженерного рекомбинантного антитела может служить любая полипептидная цепь, принадлежащая человеческому иммуноглобулину, но предпочтительно брать CH человеческих иммуноглобулинов класса G (hIgG). Кроме того, можно использовать CH любого из подклассов класса hIgG, например hIgG1, hIgG2, hIgG3 и hIgG4. CL генно-инженерного рекомбинантного антитела может служить любая полипептидная цепь, принадлежащая человеческому иммуноглобулину; можно использовать CL легкой цепи класса κ или λ.
По данному изобретению биспецифическим антителом называют полипептид или белок, специфично связывающиеся с двумя различными эпитопами. Каждый из антиген-связывающихся доменов биспецифичного антитела может связываться с отдельными эпитопами одного и того же антигена или же может связываться с различными антигенами.
В соответствии с настоящим изобретением связывание полипептида, антитела или фрагмента данного антитела, или биспецифического фрагмента данного антитела с любым антигеном клеточной поверхности, таким как EGFR или GPC3 и TfR, можно подтвердить с помощью способа, в котором аффинность связывания исследуемого антитела с клеткой которая экспрессирует антиген клеточной поверхности или TfR, подтверждается с использованием, например, известного способа иммунологического обнаружения, предпочтительно способа окрашивания клеток флуоресцентным красителем или тому подобным. Кроме того, также можно использовать известные иммунологические методы обнаружения [Monoclonal Antibodies - Principles and Practice, Third Edition, Academic Press (1996), Antibodies - A Laboratory Manual, Cold Spring Harbor Laboratory (1988), Monoclonal Antibody Experimental Manual, Kodansha scientific books (1987)] и т.п. в сочетании.
В соответствии с настоящим изобретением антигенсвязывающий домен, который связывается с антигеном клеточной поверхности, таким как EGFR или GPC3 или TfR, может быть любым, если он специфически распознает и связывается с антигеном клеточной поверхности, таким как EGFR, или GPC3, или TfR. Например, домен может быть в любой форме полипептида, белковой молекулы и ее фрагмента, конъюгата тела с низкомолекулярной молекулой или природным продуктом белковой молекулы и т.п., которое может быть получено с помощью метода рекомбинации генов, например, антитело, лиганд, рецептор или встречающаяся в природе взаимодействующая молекула.
Также антиген-связывающим доменом может служить рекомбинантный связывающий белок, в котором используется известный антиген-связывающая агент, например, антитело, лиганд или рецептор; и конкретные примеры включают рекомбинантный белок с участками CDR антитела, связывающегося с указанным антигеном, вариабельная область антитела с такими CDR, рекомбинантный белок, включающий вариабельную область антитела и связывающий домен лиганда, связывающийся с указанным антигеном, и т.п. Предпочтительно антиген-связывающим доменом по данному изобретению является вариабельная область антитела.
Примерами биспецифичного антитела или биспецифичного фрагмента антитела по данному изобретению являются биспецифические антитела или биспецифические фрагменты антител, обладающие активностью, относящейся к интернализации и/или деградации TfR. Биспецифическим антителом или биспецифическим фрагментом антитела по данному изобретению предпочтительно являются биспецифические антитела или биспецифические фрагменты антитела, не обладающие активностью, относящейся к интернализации и/или деградации TfR, в отношении клеток, в которых не экспрессируются такие антигены клеточной поверхности, как EGFR или GPC3, но проявляющие указанную активность только в отношении клеток, в которых экспрессируется такой антиген клеточной поверхности, как EGFR или GPC3. Такие биспецифические антитела или биспецифические фрагменты антител избирательно проявляют активность, относящуюся к интернализации и/или деградации TfR, в отношении патогенных клеток, например, раковых клеток, в которых экспрессируются такие антигены клеточной поверхности, как EGFR или GPC3, и поэтому такие антитела или фрагменты антител предпочтительны с точки зрения того, что не вызывают побочных эффектов, которыми сопровождаются неспецифическая интернализация и/или деградация TfR .
Под активностью, относящейся к интернализации и/или деградации TfR, которой обладают биспецифические антитела или биспецифические фрагменты антител по данному изобретению, понимается активность, результатом которой является включение TfR в процессы интернализации и/или деградации антигена клеточной поверхности при связывании на клетке указанного биспецифичного антитела или биспецифического фрагмента антитела как с антигеном клеточной поверхности, например с EGFR или GPC3, так и с TfR, что вызывает интернализацию и/или деградацию TfR .
Биспецифическое антитело или биспецифический фрагмент антитела по данному изобретению способны связываться с таким антигеном клеточной поверхности, как EGFR или GPC3, и с TfR, экспрессирующимися на одной и той же клетке, или же с таким антигеном клеточной поверхности, как EGFR или GPC3, и с TfR, экспрессирующимися на разных клетках, но предпочтительно, чтобы они связывались с антигеном клеточной поверхности, например с EGFR или GPC3, и с TfR, экспрессирующимися на одной и той же клетке.
Биспецифическое антитело или биспецифический фрагмент антитела по данному изобретению предпочтительно являются такими, что вызывают гибель клетки, в которой экспрессируется целевой антиген клеточной поверхности, такой как EGFR или GPC3, посредством чего индуцируется интернализация и/или деградация TfR .
Активность, относящаяся к интернализации и/или деградации TfR, которой обладает биспецифическое антитело или биспецифический фрагмент антитела по данному изобретению, подтверждается путем, например, определения уровня белка TfR на поверхности или внутри клеток, в которых экспрессируется TfR, например клеток OE21, T.Tn, TE-8, U-937, HepG2, HuH-7 или HLE; также можно определять количество жизнеспособных клеток, пролиферацию клеток, жизнеспособность клеток, поглощение клетками железа и проч.
Таким образом, примерами биспецифических антител или биспецифических фрагментов антител по данному изобретению являются, говоря конкретно, биспецифические антитела или биспецифические фрагменты антител, которые вызывают деградацию TfR при их связывании как с антигеном клеточной поверхности, например с EGFR или GPC3, так и с TfR, и им подобные.
Количество доменов, связывающихся с определенным антигеном, в одной молекуле биспецифического антитела - это его валентность связывания. Например, по данному изобретению, если в одной молекуле биспецифического антитела имеется два домена, связывающихся с EGFR, и два домена, связывающихся с TfR, то биспецифическое антитело связывается с каждым из EGFR и TfR бивалентно.
Биспецифические антитела по данному изобретению включают также антитела, в которых имеется множество антиген-связывающих доменов, которые соединены посредством соответствующих линкеров, включая иммуноглобулиновые домены или их фрагменты.
Биспецифические антитела по данному изобретению можно получать известными в данной области техники способами ([Nature Protocols, 9, 2450-2463 (2014), WO 1998/050431, WO 2001/7734, WO 2002/002773 и WO 2009/131239) или подобными им.
Структура биспецифического антитела по данному изобретению такова, что полипептид (в настоящем документе называемый также полипептидом N-концевой стороны), обладающий способностью связываться с антигеном клеточной поверхности, соединен непосредственно или посредством линкера с N-концом части IgG. которая связывается с TfR .
По данному изобретению иммуноглобулиновый домен включает пептид, аминокислотная последовательность которого сходна с таковой иммуноглобулина и состоит из 100 аминокислотных остатка, среди которых присутствуют по меньшей мере два остатка цистеина как наименьшая единица. Согласно данному изобретению иммуноглобулиновый домен включает также полипептид, содержащий множество иммуноглобулиновых доменов, в качестве наименьшей его единицы, описанной выше. Примеры иммуноглобулинового домена включают VH, CH1, CH2 и CH3 тяжелой цепи иммуноглобулина, а также VL и CL легкой цепи иммуноглобулина, а также подобные им.
Видовая принадлежность иммуноглобулина не ограничивается специальным образом, но предпочтительно иммуноглобулин имеет человеческое происхождение. Константная область может быть любого из подклассов IgD, IgM, IgG1, IgG2, IgG3, IgG4, IgA1, IgA2 и IgE, и предпочтительны в качестве примеров подклассы классов IgG и IgM. Кроме того, подкласс константной области легкой цепи иммуноглобулина может быть либо κ либо λ.
Также иммуноглобулиновый домен может присутствовать в белках, отличных от иммуноглобулинов; например иммуноглобулиновые домены имеются в таких представителях надсемейства иммуноглобулиновых белков, как, например, антигены главного комплекса гистосовместимости (MHC), CD1, B7 и Т-клеточный рецептор (TCR). Для биспецифических антител согласно данному изобретению можно использовать любой иммуноглобулиновый домен.
В случае человеческого IgG, CH1 называется участок с аминокислотной последовательностью с 118-го положения по 215-е положение, указанных согласно индексу EU. Аналогично, CH2 называется участок с аминокислотной последовательностью с 231-го положения по 340-е положение, указанных согласно индексу EU по Kabat et al., а CH3 оназывается участок с аминокислотной последовательностью с 341-го положения по 447-е положение , указанных согласно индексу EU по Kabat et al.. Между CH1 и CH2 расположен аминокислотный участок, обеспеченный гибкостью, также назваемый шарнирный участок (здесь и далее называемый «шарнир»). Шарнирным является участок с аминокислотной последовательностью с 216-го положения по 230-е положение , указанных согласно индексу EU по Kabat et al.
CL называется область с аминокислотной последовательностью между положениями 108 и 214, обозначенными по Кабат в случае легкой цепи типа κ человеческого антитела, и к области с аминокислотной последоватлеьностью между положениями 108 и 215 в случае легкой цепи типа λ.
Согласно данному изобретению формулировка антитело типа IgG, также относится к части IgG, и относится к части структуры IgG, входящей в состав биспецифического антитела по данному изобретению, или IgG, в котором фрагмент Fc модифицирован и имеет структуру гетеротетрамера, полученную в результате сборки двух гетеродимеров, каждый из которых состоит из одной легкой цепи и одной тяжелой цепи. Часть IgG, которая связывается с TfR, согласно » по данному изобретению – это та часть IgG, которая распознает TfR, то есть, часть IgG обладает функцией специфичного распознавания и связывания внеклеточного домена TfR.
Константная область тяжелой цепи IgG может быть любого подкласса, например, IgG1, IgG2, IgG3 или IgG4. Также часть аминокислотной последовательности этой области может быть удалена, добавлена, заменена или вставлена Кроме того, могут быть нужным образом объединены и использованы все или часть фрагментов аминокислотной последовательности, составленной из участков CH1, шарнира, CH2 и CH3 тяжелой цепи IgG. Далее, аминокислотные последовательности указанных участков могут использоваться, например, с частичными делециями или с изменением порядка их расположения. Подкласс используемой части IgG никак конкретно не ограничивается, но предпочтительны IgG4 или мутантный IgG4, полученный путем замены остатка Ser в положении 228 константной области тяжелой цепи на остаток Pro и остатка Leu в положении 235 константной области тяжелой цепи на остаток Asp (здесь и далее этот вариант обозначается IgG4PE), или мутантный IgG, полученный путем замены остатка Ser в положении 228 константной области тяжелой цепи на остаток Pro , остатка Leu в положении 235 на остаток Asp и остатка Arg в положении 409 на остаток Lys (здесь и далее этот вариант обозначается IgG4PE R409K).
К примеру, предпочтительна часть IgG, в которой константная область тяжелой цепи (CH1-шарнирный участок-CH2-CH3; указаны в порядке их расположения от N-концевой стороны) состоит из IgG4PE, содержащего аминокислотную последовательность, представленную SEQ ID NO: 86, или часть IgG, в которой CH состоит из IgG4PE R409K, содержащего аминокислотную последовательность, представленную SEQ ID NO: 84.
Две вариабельные области, входящие в состав части IgG по данному изобретению, распознают предпочтительно один и тот же антиген. Также у этих областей предпочтительно одинаковая структура и одинаковые аминокислотные последовательности.
По данному изобретению полипептидом N-концевой стороны, связывающийся с антигеном клеточной поверхности называют часть структуры биспецифического антитела по данному изобретению, а именно полипептид, расположенный на N-концевой стороне тяжелой или легкой цепи части IgG и специфично распознающий и связывающий внеклеточную область каждого антигена клеточной поверхности.
В биспецифическом антителе по данному изобретению имеются полипептиды N-концевой стороны, расположенные на N-конце тяжелой или легкой цепи в обоих гетеродимерах, составляющих часть IgG, или полипептид N-концевой стороны только на N-конце одного из гетеродимеров; однако предпочтительно, чтобы в указанном биспецифическом антителе полипептиды N-концевой стороны имелись по одному на N-конце тяжелой или легкой цепи в обоих гетеродимерах. Когда в биспецифическом антителе полипептиды N-концевой стороны имеются по одному на N-конце тяжелой или легкой цепи в обоих гетеродимерах, эти два полипептида могут быть одинаковыми либо разными, но предпочтительно они одинаковые.
Полипептид N-концевой стороны по данному изобретению может быть представлен одиночной полипептидной цепью или мультимером, состоящим из нескольких полипептидных цепей, при условии, что он обладает способностью связывать антиген клеточной поверхности. Полипептидом N-концевой стороны могут быть антиген-связывающие фрагменты антитела к антигена клеточной поверхности, а именно фрагменты Fab и Fab’, scFv, dsFv и VHH, причем предпочтительны Fab или VHH. Также в роли полипептида N-концевой стороны по данному изобретению может использоваться молекула лиганда или рецептора, специфичная к антигену клеточной поверхности.
Антитела к антигенам клеточной поверхности по данному изобретению являются моноклональными антителами, специфично распознающими и связывающими внеклеточный домен соответствующего антигена клеточной поверхности. Например, антитела к EGFR и GPC3 - это моноклональные антитела, специфично распознающие и связывающие внеклеточный домен EGFR и GPC3 соответственно.
Линкером по данному изобретению может быть любым, при условии, что он представляет собой молекулярную структуру, посредством которой соединены часть IgG и полипептид N-концевой стороны, но предпочтительно это пептидная цепь. Примерами таких линкеров являются, например, аминокислотные последовательности ES, ESKYG, ESKYGPP, GGGGS, или последовательность, состоящая из повторов GGGGS, или последовательность, представляющая собой аминокислотную последовательность шарнирного участка и константная область домена CH1, или ее часть, или что-то подобное из антитела, или подобные последовательности.
Когда в биспецифическом антителе по данному изобретению полипептидом N-концевой стороны является Fab антитела к антигену клеточной поверхности, легкая цепь в составе Fab и легкая цепь в части IgG могут быть одинаковыми либо разными, но предпочтительно они одинаковые. При этом, указанная легкая цепь может быть типа λ или κ, но предпочтительно κ.
Антитела или фрагменты антитела, которые имеют аминокислотную последовательность, являющуюся продуктом одной или более замен, добавлений, делеций или вставок аминокислотных остатков в аминокислотных последовательностях биспецифического антитела или биспецифического фрагмента антитела по данному изобретению, и которые имеют активность этого антитела или фрагмента этого антитела также входят в объем данного изобретения.
Количество удаленных, замененных, вставленных и/или добавленных аминокислотных остатков может быть равно одному или более и никак особо не ограничивается; это такое количество делеций, замен, вставок и добавлений аминокислотных остатков, которое может быть осуществлено с помощью хорощо известных в данной области техники методов, например путем сайт-специфический мутагенеза, описанных в Molecular Cloning, The Second Edition, Cold Spring Harbor Laboratory Press (1989), Current Protocols in Molecular Biology, John Willy & Sons (1987-1997), Nucleic Acids Research, 10, 6487 (1982), Proc. Natl. Acad. Sci., USA, 79, 6409 (1982), Gene, 34, 315 (1985), Nucleic Acids Research, 13, 4431 (1985), Proc. Natl. Acad. Sci USA, 82, 488 (1985), и др. Например. это количество может составлять от 1 до нескольких десятков, предпочтительно 1-20, более предпочтительно 1-10 и еще более предпочтительно 1-5.
Приведенное выше описание, что один или несколько аминокислотных остатков в аминокислотной последовательности биспецифического антитела настоящего изобретения могут быть удалены, заменены, вставлены или добавлены означает следующее. Это описание означает, что произошла делеция, замена, вставка или добавление одного или множества аминокислотных остатков в произвольной одном или нескольких местах одной последовательности. Далее подобные делеции, замены, вставки или добавления аминокислотных остатков иногда происходят одновременно, причем заменяемые, добавляемые или вставляемые аминокислотные остатки могут быть такими, какие встречаются в природных белках, или же не встречающимися в природе.
Примеры природных аминокислот включают L-аланин, L-аспарагин, L-аспарагиновую кислоту, L-глутамин, глутаминовую кислоту, глицин, L-гистидин, L-изолейцин, L-лейцин, L-лизин, L-аргинин, L-метионин, L-фенилаланин, L-пролин, L-серин, L-треонин, L-триптофан, L-тирозин, L-валин, L-цистеин и др.
Далее в настоящем документе приводятся предпочтительные примеры взаимозаменяемых аминокислотных остатков. Остатки аминокислот, относящихся к одной и той же группе из указанных ниже, являются взаимозаменяемыми.
Группа A: лейцин, изолейцин, норлейцин, валин, норвалин, аланин, 2-аминомасляная кислота, метионин, О-метилсерин, трет-бутилглицин, трет-бутилаланин и циклогексилаланин.
Группа B: аспарагиновая кислота, глутаминовая кислота, изоаспарагиновая кислота, изоглутаминовая кислота, 2-аминоадипиновая кислота и 2-аминосубериновая кислота.
Группа C: аспарагин и глутамин.
Группа D: лизин, аргинин, орнитин, 2,4-диаминомасляная кислота и 2,3-диаминопропионовая кислота.
Группа E: пролин, 3-гидроксипролин и 4-гидроксипролин.
Группа F: серин, треонин и гомосерин.
Группа G: фенилаланин и тирозин.
Биспецифическое антитело и бсспецифический фрагмент этого антитела по данному изобретению включают также антитело, содержащее какие-либо аминокислотные остатки, подвергшиеся посттрансляционной модификации. Примеры посттрансляционной модификации включают делецию остатка Lys на С-конце цепи H (отщепление Lys), замену остатка глутамина на N-конце полипептидной цепи на пироглутамат (pyroGlu) и др. [Beck et al, Analytical Chemistry, 85, 715-736 (2013)].
Конкретные примеры биспецифических антител по данному изобретению включают любое биспецифическое антитело, выбираемое из группы, состоящей из указанных ниже (1)-(4), и подобных им:
(1) биспецифическое антитело, включающее Fab, который содержит вариабельную область антитела к антигену клеточной поверхности, и часть IgG, связывающуюся с TfR, причем Fab непосредственно соединен с N-концом тяжелой цепи части IgG;
(2) биспецифическое антитело, включающее Fab, который содержит VH, имеющий CDR 1-3 из VH, и VL, имеющий CDR 1-3 из VH, причем указанные VH и VL происходят из антитела к антигену клеточной поверхности, и часть IgG, связывающуюся с TfR, причем Fab непосредственно соединен с N-концом тяжелой цепи части IgG;
(3) биспецифическое антитело, включающее Fab, который содержит VH и VL антитела к антигену клеточной поверхности, и часть IgG, связывающуюся с TfR, причем Fab непосредственно соединен с N-концом тяжелой цепи части IgG; и
(4) биспецифическое антитело, включающее VHH, который связывается с антигеном клеточной поверхности, и часть IgG, связывающуюся с TfR, причем VHH непосредственно соединен с N-концом тяжелой цепи части IgG;
В биспецифических антителах, описанных выше в пунктах (1)-(3), VL в части IgG, связывающейся с TfR, и VL Fab могут быть одинаковыми либо разными, но предпочтительно они одинаковые.
Примером части IgG, связывающейся с TfR, по данному изобретению является часть IgG, которая связывается с TfR и включает VL, содержащую CDR 1, 2 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NOS: 17, 18 и 19 соответственно, и VH, содержащую CDR 1-3, примеры которых приведены ниже в пунктах 1) - 3).
1) CDR1, содержащий аминокислотную последовательность, представленную SEQ ID NO: 32, или продукт модификации аминокислотной последовательности, представленной SEQ ID NO: 32, путем по меньшей мере одной замены, выбираемой из замены тирозина в положении 2 на аланин, или фенилаланин и замены треонина в положении 3 на аланин или глицин.
2) CDR2, содержащий аминокислотную последовательность, представленную SEQ ID NO: 33, или продукт модификации аминокислотной последовательности, представленной SEQ ID NO: 33, путем по меньшей мере одной замены , выбираемой из замен валина в положении 1 на аланин, изолейцина в положении 2 на аланин или лейцин, валина в положении 7 на глутаминовую кислоту, аспарагиновой кислоты в положении 10 на аланин и аспарагиновой кислоты в положении 13 на пролин.
3) CDR3, содержащий аминокислотную последовательность, представленную SEQ ID NO: 34, или продукт модификации аминокислотной последовательности, представленной SEQ ID NO: 34, путем по меньшей мере одной замены , выбираемой из замен глутамина в положении 3 на аланин или аспарагиновую кислоту, пролина в положении 4 на произвольно выбранную природную аминокислоту, триптофана в положении 5 на аланин, фенилаланин, гистидин или тирозин, тирозина в положении 7 на аланин или фенилаланин и валина в положении 13 на лейцин. При замене пролина в положении 4 на произвольно выбранную природную аминокислоту примерами этой последней являются аланин, тирозин, серин, аспарагиновая кислота, глутамин, глутаминовая кислота, треонин, аргинин, глицин, лизин, метионин, валин, лейцин, изолейцин, триптофан, фенилаланин или гистидин.
Часть IgG, связывающаяся с TfR, по данному изобретению включает также часть IgG, связывающуюся с TfR и содержащую аминокислотные последовательности CDR 1-3 VH, и VL, имеющие по меньшей мере на 35%, по меньшей мере на 40%, по меньшей мере на 45%, по меньшей мере на 50%, по меньшей мере на 55%, по меньшей мере на 60%, по меньшей мере на 65%, по меньшей мере на 70%, по меньшей мере на 75%, по меньшей мере на 80%, по меньшей мере на 85%, по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% гомологичности с соответствующими аминокислотными последовательностями CDR 1-3 VH, представленными SEQ ID NOS: 32, 33 и 34 соответственно, и аминокислотным последовательностям CDR 1-3 VL, представленными SEQ ID NOS: 17 – 19 соответственно.
Часть IgG, связывающаяся с TfR, по данному изобретению включает также антитела, описанные ниже в пункте (ai) или (aii)
(ai) Часть IgG, связывающаяся с TfR, которая конкурирует за связывание с TfR с анти- TfR антителом к, включающим VL с участками CDR 1, 2 и 3, содержащими аминокислотные последовательности, представленные SEQ ID NО: 17, 18 и 19 соответственно, и VH с участками CDR 1, 2 и 3, содержащими аминокислотные последовательности, представленные SEQ ID NOS: 32, 33, и 34 соответственно; или
(aii) Часть IgG, связывающаяся с TfR и взаимодействующая с тем же эпитопом, что и антитело к TfR, включающее VL с CDR 1, 2 и 3, содержащими аминокислотные последовательности, представленные SEQ ID NО: 17, 18 и 19 соответственно, и VH с CDR 1, 2 и 3, содержащими аминокислотные последовательности, представленные SEQ ID NOS: 32, 33, и 34 соответственно.
Часть IgG, связывающаяся с TfR, по данному изобретению включает также антитела, описанные ниже в пункте (bi) или (bii):
(bi) часть IgG, конкурирующая за связывание с TfR с антителом к TfR, включающим VL с CDR 1, 2 и 3, содержащими аминокислотные последовательности, представленные SEQ ID NО: 17, 18 и 19 соответственно, и VH с CDR 1, 2 и 3, содержащими аминокислотные последовательности, представленные SEQ ID NО: 42, 43 и 44 соответственно; или
(bii) часть IgG, которая связывается с TfR, взаимодействуя с тем же эпитопом, что и антитело к TfR, включающее VL с CDR 1, 2 и 3, содержащими аминокислотные последовательности, представленные SEQ ID NО: 17, 18 и 19 соответственно, и VH с CDR 1, 2 и 3, содержащими аминокислотные последовательности, представленные SEQ ID NО: 42, 43 и 44 соответственно.
Часть IgG, связывающаяся с TfR, по данному изобретению включает также антитела, описанные ниже в пункте (ci) или (cii):
(ci) часть IgG, конкурирующая за связывание с TfR с антителом к TfR, включающим VL с CDR 1, 2 и 3, содержащими аминокислотные последовательности, представленные SEQ ID NО: 17, 18 и 19 соответственно, и VH с CDR 1, 2 и 3, содержащими аминокислотные последовательности, представленные SEQ ID NО: 47, 48 и 49 соответственно; или
(cii) часть IgG, которая связывается с TfR, взаимодействуя с тем же эпитопом, что и антитело к TfR, включающее VL с CDR 1, 2 и 3, содержащими аминокислотные последовательности, представленные SEQ ID NО: 17, 18 и 19 соответственно, и VH с CDR 1, 2 и 3, содержащими аминокислотные последовательности, представленные SEQ ID NО: 47, 48 и 49 соответственно.
Другим примером части IgG, которая связывается с TfR, по данному изобретению является часть IgG, связывающаяся с TfR и содержащая VL, включающую аминокислотную последовательность, представленную SEQ ID NO: 16, и VH, включающую аминокислотную последовательность, представленную SEQ ID NO: 26, 31, 36, 41, 46 или 106.
Одним из примеров части IgG, которая связывается с TfR, по данному изобретению является часть IgG, связывающаяся с TfR и содержащая VL с CDR 1-3, содержащими аминокислотные последовательности, представленные SEQ ID NО: 17-19 соответственно, и VH, выбираемую из следующего:
(a) VH, включающая CDR 2 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 33 и 34 соответственно, и CDR1, содержащий аминокислотную последовательность, являющуюся продуктом модификации аминокислотной последовательности, представленной SEQ ID NO: 32, путем замены тирозина в положении 2 на аланин или фенилаланин;
(b) VH, включающая CDR 2 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 33 и 34 соответственно, и CDR1, содержащий аминокислотную последовательность, являющуюся продуктом модификации аминокислотной последовательности, представленной SEQ ID NO: 32, путем замены треонина в положении 3 на аланин или глицин;
(c) VH, включающая CDR 1 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 32 и 34 соответственно, и CDR2, содержащий аминокислотную последовательность, являющуюся продуктом модификации аминокислотной последовательности, представленной SEQ ID NO: 33, путем замены валина в положении 1 на аланин;
(d) VH, включающая CDR 1 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 32 и 34 соответственно, и CDR2, содержащий аминокислотную последовательность, являющуюся продуктом модификации аминокислотной последовательности, представленной SEQ ID NO: 33, путем замены изолейцина в положении 2 на аланин или лейцин;
(e) VH, включающая CDR 1 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 32 и 34 соответственно, и CDR2, содержащий аминокислотную последовательность, являющуюся продуктом модификации аминокислотной последовательности, представленной SEQ ID NO: 33, путем замены валина в положении 7 на глутаминовую кислоту;
(f) VH, включающая CDR 1 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 32 и 34 соответственно, и CDR2, содержащий аминокислотную последовательность, являющуюся продуктом модификации аминокислотной последовательности, представленной SEQ ID NO: 33, путем замены аспарагиновой кислоты в положении 10 на аланин;
(g) VH, включающая CDR 1 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 32 и 34 соответственно, и CDR2, содержащий аминокислотную последовательность, являющуюся продуктом модификации аминокислотной последовательности, представленной SEQ ID NO: 33, путем замены аспарагиновой кислоты в положении 13 на пролин;
(h) VH, включающая CDR 1 и 2, содержащие аминокислотные последовательности, представленные SEQ ID NO: 32 и 33 соответственно, и CDR3, содержащий аминокислотную последовательность, являющуюся продуктом модификации аминокислотной последовательности, представленной SEQ ID NO: 34, путем замены глутамина в положении 3 на аланин или аспарагиновую кислоту;
(i) VH, включающая CDR 1 и 2, содержащие аминокислотные последовательности, представленные SEQ ID NO: 32 и 33 соответственно, и CDR3, содержащий аминокислотную последовательность, являющуюся продуктом модификации аминокислотной последовательности, представленной SEQ ID NO: 34, путем замены пролина в положении 4 на произвольно выбранную аминокислоту;
(j) VH, включающая CDR 1 и 2, содержащие аминокислотные последовательности, представленные SEQ ID NO: 32 и 33 соответственно, и CDR3, содержащий аминокислотную последовательность, являющуюся продуктом модификации аминокислотной последовательности, представленной SEQ ID NO: 34, путем замены пролина в положении 4 на аланин, тирозин, серин, аспарагиновую кислоту, глутамин, глутаминовую кислоту, треонин, аргинин, глицин, лизин, метионин, валин, лейцин, изолейцин, триптофан, фенилаланин или гистидин;
(k) VH, включающая CDR 1 и 2, содержащие аминокислотные последовательности, представленные SEQ ID NO: 32 и 33 соответственно, и CDR3, содержащий аминокислотную последовательность, являющуюся продуктом модификации аминокислотной последовательности, представленной SEQ ID NO: 34, путем замены триптофана в положении 5 на аланин, фенилаланин, гистидин или тирозин;
(l) VH, включающая CDR 1 и 2, содержащие аминокислотные последовательности, представленные SEQ ID NO: 32 и 33 соответственно, и CDR3, содержащий аминокислотную последовательность, являющуюся продуктом модификации аминокислотной последовательности, представленной SEQ ID NO: 34, путем замены тирозина в положении 7 на аланин или фенилаланин; и
(m) VH, включающая CDR 1 и 2, содержащие аминокислотные последовательности, представленные SEQ ID NO: 32 и 33 соответственно, и CDR3, содержащий аминокислотную последовательность, являющуюся продуктом модификации аминокислотной последовательности, представленной SEQ ID NO: 34, путем замены валина на лейцин.
Одним из примеров части IgG, которая связывается с TfR, по данному изобретению является часть IgG, связывающаяся с TfR и включающая VL, содержащую аминокислотную последовательность, представленную SEQ ID NO: 16, и VH, содержащую аминокислотную последовательность, являющуюся продуктом модификации аминокислотной последовательности, представленной SEQ ID NO: 31, путем по меньшей мере одной замены, выбираемой из Y32A, Y32F, T33A, T33G, L45A, V48A, V50A, I51A, I51L, V55E, D58A, D61P, Q97A, Q97D, W99A, W99F, W99H, W99Y, Y100AA, Y100AF, V102L и P98 на произвольно выбранную аминокислоту, как определено по индексу EU, как Kabat et al. (здесь и далее индекс EU).
Одним из примеров части IgG, которая связывается с TfR, по данному изобретению является часть IgG, связывающаяся с TfR и включающая VL, содержащую аминокислотную последовательность, представленную SEQ ID NO: 16, и VH, содержащую аминокислотную последовательность, являющуюся продуктом модификации аминокислотной последовательности, представленной SEQ ID NO: 31, путем по меньшей мере одной замены, выбираемой из Y32A, Y32F, T33A, T33G, L45A, V48A, V50A, I51A, I51L, V55E, D58A, D61P, Q97A, Q97D, W99A, W99F, W99H, W99Y, Y100AA, Y100AF, V102L, P98A, P98Y, P98S, P98D, P98Q, P98E, P98T, P98R, P98G, P98K, P98M, P98V, P98L, P98I, P98W, P98F и P98H, как определено по индексу EU.
Одним из примеров части IgG, которая связывается с TfR, по данному изобретению является часть IgG, связывающаяся с TfR и распознающая по меньшей мере один аминокислотный остаток, выбираемый из остатков Asp в положении 352, Ser в положении 355, Asp в положении 356 и Lys в положении 358, и по меньшей мере один аминокислотный остаток, выбираемый из остатков Met в положении 365, Val в положении 366 и Glu в положении 369 аминокислотной последовательности TfR, представленной SEQ ID NO: 6. В одном из воплощений данного изобретения частью IgG, которая связывается с TfR, является, например, часть IgG, связывающаяся с TfR и распознающая по меньшей мере два аминокислотных остатка, выбираемых из остатков Asp в положении 352, Ser в положении 355, Asp в положении 356 и Lys в положении 358, и по меньшей мере два аминокислотных остатка, выбираемых из остатков Met в положении 365, Val в положении 366 и Glu в положении 369; часть IgG, связывающаяся с TfR и распознающая по меньшей мере три аминокислотных остатка, выбираемых из остатков Asp в положении 352, Ser в положении 355, Asp в положении 356 и Lys в положении 358, и все следующие аминокислотные остатки: Met в положении 365, Val в положении 366 и Glu в положении 369; и часть IgG, связывающаяся с TfR и распознающая все следующие аминокислотные остатки: Asp в положении 352, Ser в положении 355, Asp в положении 356 и Lys в положении 358, - и все следующие аминокислотные остатки: Met в положении 365, Val в положении 366 и Glu в положении 369.
Одним из примеров анти-EGFR антитела по данному изобретению , является антитело к EGFR, в котором имеются VL, содержащая CDR 1, 2 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NО: 17, 18 и 19 соответственно, и любая из вариабельных областей тяжелой цепи (VH), выбираемая из приведенных ниже в пунктах (2a)-(2b).
(2a) VH, включающая CDR 1, 2 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NО: 60, 61 и 62 соответственно; и
(2b) VH, включающая CDR 1, 2 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NО: 65, 66 и 67 соответственно.
Антитело к EGFR по данному изобретению включает также антитела, которые содержат аминокислотные последовательности CDR 1-3 VH и VL, по меньшей мере на 35%, по меньшей мере на 40%, по меньшей мере на 45%, по меньшей мере на 50%, по меньшей мере на 55%, по меньшей мере на 60%, по меньшей мере на 65%, по меньшей мере на 70%, по меньшей мере на 75%, по меньшей мере на 80%, по меньшей мере на 85%, по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% гомологичные соответствующим аминокислотным последовательностям CDR 1 -3 VH, описанной в приведенных выше пунктах (2a) и (2b), и аминокислотным последовательностям CDR 1 -3 VL, представленным SEQ ID NO: 17 - 19 соответственно.
Антитело к EGFR по данному изобретению включает также антитела, описанные в приведенном ниже пункте (i) или (ii):
(i) антитело, связывающее EGFR конкурентно с антителом к EGFR, в котором имеются VL, включающая CDR 1, 2 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 17, 18 и 19 соответственно, и VH, описанная любым из приведенных выше пунктов (2a) - (2b); или
(ii) антитело, связывающее тот же эпитоп, что и антитело к EGFR, в котором имеются VL, включающая CDR 1, 2 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 17, 18 и 19 соответственно, и VH, описанная любым из приведенных выше пунктов (2a) - (2b).
Другим примером антитела к EGFR по данному изобретению является биспецифическое антитело, выбираемое из антител, описанных в приведенных ниже пунктах (a) - (e):
(a) антитело, включающее VH, содержащую аминокислотную последовательность, представленную SEQ ID NO: 59 или SEQ ID NO: 64, и VL, содержащую аминокислотную последовательность, представленную SEQ ID NO: 16;
(b) антитело, включающее VH, содержащую аминокислотную последовательность, представленную SEQ ID NO: 110, и VL цепи, содержащую аминокислотную последовательность, представленную SEQ ID NO: 111;
(c) антитело, включающее VH, содержащую аминокислотную последовательность, представленную SEQ ID NO: 112, и VL, содержащую аминокислотную последовательность, представленную SEQ ID NO: 113;
(d) антитело, включающее VH, содержащую аминокислотную последовательность, представленную SEQ ID NO: 114, и VL, содержащую аминокислотную последовательность, представленную SEQ ID NO: 115; и
(e) антитело, включающее VH, содержащую аминокислотную последовательность, представленную SEQ ID NO: 116, и VL, содержащую аминокислотную последовательность, представленную SEQ ID NO: 117.
В одном из воплощений данного изобретения примером биспецифического антитела является антитело, которое включает полипептид N-концевой стороны и часть IgG, связывающуюся с TfR, и в котором полипептид N-концевой стороны представлен фрагментом Fab, причем С-конец участка VH-CH1 Fab соединен непосредственно или посредством линкера с N-концом части IgG, связывающейся с TfR.
Также в одном из воплощений данного изобретения примером биспецифического антитела является антитело, которое включает полипептид N-концевой стороны и часть IgG, связывающуюся с TfR, и в котором полипептид N-концевой стороны является фрагментом Fab, причем С-конец VL-СL области Fab соединен непосредственно или посредством линкера с N-концом тяжелой цепи части IgG, связывающейся с TfR.
В одном из воплощений данного изобретения примером биспецифического антитела является антитело, которое включает часть IgG, связывающуюся с TfR, и полипептид N-концевой стороны, связывающийся с EGFR.
Более конкретные примеры биспецифических антител по данному изобретению включают биспецифические антитела, описанные в приведенных ниже пунктах (i) - (iv).
(i) Биспецифическое антитело, включающее Fab антитела к EGFR, в котором имеются VL, включающая CDR 1, 2 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NOS: 17, 18 и 19 соответственно, и VH, включающая CDR 1, 2 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 60, 61 и 62 соответственно, и включающее часть IgG, связывающую TfR , в которой имеются VL, включающая CDR 1, 2 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NOS: 17, 18 и 19 соответственно, и какая-либо VH, выбираемая из описанных в приведенных ниже пунктах (a) - (f), в котором Fab соединен непосредственно или через линкер с N-концом тяжелой цепи части IgG, связывающейся с TfR :
(a) VH, включающая CDR 1, 2 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 22, 23 и 24 соответственно;
(b) VH, включающая CDR 1, 2 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 27, 28, и 29 соответственно;
(c) VH, включающая CDR 1, 2 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 32, 33, и 34 соответственно;
(d) VH, включающая CDR 1, 2 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 37, 38 и 39 соответственно;
(e) VH, включающая CDR 1, 2 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 42, 43 и 44 соответственно; и
(f) VH, включающая CDR 1, 2 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 47, 48 и 49 соответственно.
(ii) Биспецифическое антитело, включающее Fab антитела к EGFR, в котором имеются VL, включающая CDR 1, 2 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 17, 18 и 19 соответственно, и VH, включающая CDR 1, 2 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NО: 65, 66 и 67 соответственно, и включающее часть IgG, связывающуюся с TfR, которая содержит VL, включающую CDR 1, 2 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NOS: 17, 18 и 19 соответственно, и какая-либо VH, выбираемая из описанных в приведенных ниже пунктах (a)-(f), в котором указанный Fab соединен непосредственно или через линкер с N-концом тяжелой цепи части Ig, связывающейся с TfR :
(a) VH, включающая CDR 1, 2 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 22, 23 и 24 соответственно;
(b) VH, включающая CDR 1, 2 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 27, 28 и 29 соответственно;
(c) VH, включающая CDR 1, 2 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 32, 33 и 34 соответственно;
(d) VH, включающая CDR 1, 2 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 37, 38 и 39 соответственно;
(e) VH, включающая CDR 1, 2 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 42, 43 и 44 соответственно; и
(f) VH, включающая CDR 1, 2 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 47, 48 и 49 соответственно.
(iii) Биспецифическое антитело, включающее Fab антитела к EGFR, в котором имеются VL, включающая CDR 1, 2 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 17, 18 и 19 соответственно, и VH, включающая CDR 1, 2 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NО: 70, 71 и 72 соответственно, и включающее часть IgG, связывающуюся с TfR, в которой имеются VL, включающая CDR 1, 2 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NOS: 17, 18 и 19 соответственно, иVH, выбираемая из описанных в приведенных ниже пунктах (a) - (f), в котором указанный Fab соединен непосредственно или через линкер с N-концом тяжелой цепи части Ig, связывающейся с TfR :
(a) VH, включающая CDR 1, 2 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 22, 23 и 24 соответственно;
(b) VH, включающая CDR 1, 2 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 27, 28 и 29 соответственно;
(c) VH, включающая CDR 1, 2 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 32, 33 и 34 соответственно;
(d) VH, включающая CDR 1, 2 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 37, 38 и 39 соответственно;
(e) VH, включающая CDR 1, 2 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 42, 43 и 44 соответственно; и
(f) VH, включающая CDR 1, 2 и 3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 47, 48 и 49 соответственно.
(iv) Биспецифическое антитело, включающее Fab антитела к EGFR, в котором имеются VL, содержащая аминокислотную последовательность, представленную SEQ ID NO: 16, и VH, содержащая аминокислотную последовательность, представленную SEQ ID NO: 59, и включающее часть IgG, связывающуюся с TfR, в которой имеются VL, включающая аминокислотную последовательность, представленную SEQ ID NO: 16, и VH, включающая аминокислотную последовательность, представленную SEQ ID NO: 21, 26, 31, 36, 41 или 46, в котором указанный Fab соединен непосредственно или через линкер с N-концом тяжелой цепи части Ig, связывающейся с TfR .
(v) Биспецифическое антитело, включающее Fab антитела к EGFR, в котором имеются VL, содержащая аминокислотную последовательность, представленную SEQ ID NO: 16, и VH, содержащая аминокислотную последовательность, представленную SEQ ID NO: 64, и включающее часть IgG, связывающуюся с TfR, в которой имеются VL, включающая аминокислотную последовательность, представленную SEQ ID NO: 16, и VH, включающая аминокислотную последовательность, представленную SEQ ID NO: 21, 26, 31, 36, 41 или 46, в котором указанный Fab соединен непосредственно или через линкер с N-концом тяжелой цепи части Ig, связывающейся с TfR .
(vi) Биспецифическое антитело, включающее Fab антитела к EGFR, в котором имеются VL, содержащая аминокислотную последовательность, представленную SEQ ID NO: 16, и VH, содержащая аминокислотную последовательность, представленную SEQ ID NO: 69, и включающее часть IgG, связывающуюся с TfR, в которой имеются VL, включающая аминокислотную последовательность, представленную SEQ ID NO: 16, и VH, включающая аминокислотную последовательность, представленную SEQ ID NO: 21, 26, 31, 36, 41 или 46, в котором указанный Fab соединен непосредственно или через линкер с N-концом тяжелой цепи части Ig, связывающейся с TfR .
В биспецифическом антителе, описанном в приведенных выше пунктах (i) - (vi), областью, соединенной непосредственно или посредством линкера с N-концом тяжелой цепи части IgG, связывающейся с TfR, может быть область VH-CH1 либо VL-CL из Fab, но предпочтительно это VH-CH1.
Примером одного из воплощений данного изобретения является биспецифическое антитело, в котором полипептид N-концевой стороны соединен непосредственно или посредством линкера с N-концом тяжелой цепи части IgG, связывающейся с TfR, и в котором константная область части IgG, связывающейся с TfR, принадлежит к подтипу IgG1, или IgG4, или их модификациям. Примером более предпочтительного воплощения данного изобретения является биспецифическое антитело, которое включает IgG4PE или IgG4PE R409K в константной области тяжелой цепи части IgG, связывающейся с TfR, и в котором полипептид N-концевой стороны соединен непосредственно или посредством линкера с N-концом тяжелой цепи части IgG, связывающейся с TfR .
Примером еще одного из воплощений данного изобретения является биспецифическое антитело, которое включает часть IgG, связывающуюся с TfR, и полипептид N-концевой стороны, связывающийся с рецептором EGFR, и в котором полипептид N-концевой стороны является фрагментом Fab, который соединен с N-концом тяжелой цепи указанной части IgG посредством линкера. Примером более предпочтительного воплощения данного изобретения является биспецифическое антитело, в котором линкер имеет аминокислотную последовательность, выбираемую из последовательностей ES, ESKYG, ESKYGPP, GGGGS или последовательности из повторов GGGGS.
Примером биспецифического антитела по данному изобретению являются антитела E08-TfR1071 или E12-TfR1071.
Биспецифическое антитело или биспецифический фрагмент этого антитела по данному изобретению включают также антитело или фрагмент антитела , обладающие эффекторной активностью.
Под эффекторной активностью по данному изобретению понимается активность антитело-зависимой клеточной цитотоксичности, которая опосредуется областью Fc молекулы антитела; примеры этой активности включают антитело-зависимую клеточную цитотоксичность (активность ADCC), цитотоксичность, зависимую от комплемента (активность CDC), антитело-зависимый клеточный фагоцитоз (активность ADCP), происходящий при участии фагоцитирующих клеток (например, макрофагов или дендритных клеток), опсонизация и др.
По данному изобретению активности ADCC и CDC количественно определяются с помощью известных аналитических методов [Cancer Immunol. Immunother., 36, 373 (1993)].
Активностью ADCC называют активность, которая активирует иммунную клетку (естветственную киллерную клетку или подобную ей), когда антитело, связавшее антиген на клетке-мишени, своей областью Fc связывается с рецептором Fc иммунной клетки, в результате чего целевая клетка повреждается.
Рецептор Fc (FcR) – это рецептор, который связывается с областью Fc антитела и вызывает различные эффекторные активности антитела. Каждому подклассу антител соответствует свой FcR, и IgG, IgE, IgA и IgM специфически связываются с FcγR, FcγR, FcγR и FcγR соответственно. Кроме того, у FcγR есть подтипы FcγRI (CD64), FcγRII (CD32) и FcγRIII (CD16), и подтипы имеют изоформы FcγRIA, FcγRIB, FcγRIC, FcγRIIA, FcγRIIB, FcγRIIC, FcγRIIIA и FcγRIIIB соответственно. Различные типы FcγR присутствуют в разных клетках [Annu. Rev. Immunol. 9: 457-492 (1991)]. У людей FcγRIIIB специфически экспрессируется в нейтрофилах, а FcγRIIIA экспрессируется в моноцитах, естественных клетках-киллерах (NK-клетках), макрофагах и некоторых Т-клетках. Активность ADCC, зависимая от NK-клеток, индуцируется связыванием антитела с FcγRIIIA.
Активностью CDC называют активность, которая активирует серию каскадов (путей активации комплемента), состоящих из групп белков, связанных с комплементом, в крови с помощью антитела, связавшегося с антигеном на клетке-мишени, чтобы повредить клетку-мишень. Кроме того, фрагмент белка, образующийся при активации комплемента, вызывает миграцию и активацию иммунной клетки. Каскад активности CDC начинается, когда C1q сначала связывается с областью Fc, а затем связывается с C1r и C1s, которые являются двумя сериновыми протеазами, в результате чего образуется комплекс C1.
Активность CDC или активность ADCC биспецифического антитела или его фрагмента по настоящему изобретению против антиген-экспрессирующей клетки можно оценить известным методом измерения [Cancer Immunol. Immunother., 36, 373 (1993)].
В качестве способа контроля эффекторной активности биспецифического антитела по настоящему изобретению известны способ контроля количества фукозы (также называемой ядром фукозы), которая представляет собой α-1,6-связанную с N-ацетилглюкозамином (GlcNAc), присутствующим на восстанавливающем конце N-связанной сложной сахарной цепи, которая связывается с аспарагином (Asn) в положении 297 области Fc (константная область, состоящая из доменов CH2 и CH3) антитела (WO 2005/035586, WO 2002/31140 и WO 00/61739), способ контроля эффекторной активности путем модификации аминокислотного остатка Fc-области антитела (WO 00/42072) и т.п.
ADCC-активность антитела может быть увеличена или уменьшена путем регулирования количества фукозы, добавляемой к биспецифическому антителу. Например, в качестве способа уменьшения содержания фукозы, которая связывается с N-связанной сложной сахарной цепью, связанной с Fc антитела, может быть использована экспрессия биспецифического антитела с использованием клетки-хозяина, дефицитной по гену α1,6-фукозилтрансферазы, в результате чего может быть получено антитело с высокой ADCC. С другой стороны, в качестве способа увеличения содержания фукозы, которая связывается с N-связанной сложной сахарной цепью, связанной с Fc биспецифического антитела, может быть использована экспрессия антитела с использованием клетки-хозяина, трансфицированной геном α1,6-фукозилтрансферазы, в результате чего можно получить биспецифическое антитело, имеющее низкую активность ADCC.
Кроме того, активность ADCC или активность CDC может быть увеличена или уменьшена путем модификации аминокислотного остатка в области Fc биспецифического антитела. Например, используя аминокислотную последовательность области Fc, описанную в публикации заявки на патент США № 2007/0148165, можно повысить активность CDC биспецифического антитела. Кроме того, выполняя модификацию аминокислот, описанную в патенте США № 6737056, патенте США № 7297775, патенте США № 7317091 и т.п., активность ADCC или активность CDC может быть увеличена или уменьшена.
Кроме того, биспецифическое антитело, в котором контролируется эффекторная активность, может быть получено путем комбинирования вышеупомянутых методов.
Стабильность биспецифических антител по данному изобретению можно оценить, измеряя количество агрегатов (олигомеров антител), образующихся в образце в процессе очистки и хранения или в определенных условиях. Если в одних и тех же условиях количество агрегатов стало меньше, значит стабильность антитела улучшилась. Количество агрегатов можно определить, отделив агрегированные антитела от не агрегированных с помощью хроматографических методов, в том числе путем гель-фильтрационной хроматографии.
Выход биспецифического антитела по настоящему изобретению можно оценить путем измерения количества антитела, продуцируемого клеткой, продуцирующей антитело, в культуральном растворе. Более конкретно, продуктивность может быть оценена путем измерения количества антитела, содержащегося в культуральном супернатанте, полученном путем удаления продуцирующей клетки из культурального раствора с использованием подходящего метода, такого как метод ВЭЖХ или метод ELISA.
В настоящем изобретении фрагмент антитела представляет собой белок, который включает антигенсвязывающий домен и обладает антигенсвязывающей активностью по отношению к антигену. Например, Fab, Fab’, F(ab’)2, scFv, диатело, dsFv, VHH, пептид, включающий CDR или тому подобное.
Fab представляет собой фрагмент антитела, который имеет молекулярную массу около 50000 и обладает антигенсвязывающей активностью, и в котором примерно половина цепи H на N-конце и полная цепь L связаны дисульфидной связью (SS-связь), получаемый при обработке антитела IgG протеазой папаином (с расщеплением по аминокислотному остатку в положении 224 в цепи H). Цепь H из Fab включает VH и CH1 и обозначается как VH-CH1. При этом, цепь L из Fab включает VL и CL и называется VL-CL.
F (ab')2 представляет собой фрагмент антитела, который имеет молекулярную массу около 100000, обладает антигенсвязывающей активностью, и он немного больше, чем молекула, полученная путем связывания Fab через связь S-S в шарнирной области, , получаемый обработкой IgG протеазой пепсином (с расщеплением по аминокислотному остатку в положении 234 в цепи H).
Fab’ представляет собой фрагмент антитела, который имеет молекулярную массу около 50 000 и обладает антигенсвязывающей активностью, и в котором связь S-S в шарнирной области указанного выше F (ab’) 2 расщеплена.
ScFv представляет собой полипептид VH-P-VL или VL-P-VH, в котором один VH и один VL связаны с использованием соответствующего пептидного линкера (P) из 12 или более остатков, и представляет собой фрагмент антитела, обладающий антигенсвязывающей активностью.
Диатело представляет собой фрагмент антитела, в котором scFv, имеющие одинаковую или разную антигенсвязывающую специфичность, образуют димер, и представляет собой фрагмент антитела, обладающий двухвалентной антигенсвязывающей активностью по отношению к одному и тому же антигену или антигенсвязывающей активностью, каждый из которых специфичен для разных антигенов.
dsFv относится к молекуле, полученной связыванием полипептидов, в которых один аминокислотный остаток в каждом из VH и VL замещен остатком цистеина через связь S-S между остатками цистеина.
VHH (также называемый нанотелом) - это вариабельная область тяжелой цепи в антителе VHH и может связываться с антигеном без присутствия другого полипептида.
Антитело VHH представляет собой антитело, присутствующее у животного семейства Camelidae, такого как альпака, и у эластожаберных, таких как акула, и не включает легкую цепь или CH1, и состоит только из тяжелой цепи.
Пептид, включающий CDR, имеет такую конфигурацию, чтобы содержать по меньшей мере один участок CDR VH или VL. Можно получить пептид, включающий множество CDR, путем присоединения CDR непосредственно или посредством подходящего пептидного линкера. Для получения пептида, включающего CDR конструируют ДНК, кодирующие CDR VH и VL биспецифического антитела по данному изобретению, встраивают эту ДНК в экспрессионный вектор для прокариотических либо эукариотических клеток и вводят его в соответствующие клетки, что приводит к экспрессии. Также пептид, включающий CDR, может быть получен путем химического синтеза с использованием фторенилметилоксикарбонила (Fmoc) или трет-бутилоксикарбонила (tBoc).
В настоящем изобретении биспецифический фрагмент антитела по существу состоит из части структуры биспецифического антитела и представляет собой фрагмент биспецифического антитела, обладающий антигенсвязывающей активностью по отношению к двум типам антигенов.
Слитый белок, в котором связаны Fc биспецифического антитела по настоящему изобретению и фрагмент антитела, слитый белок Fc (также называемый иммуноадгезином), в котором связаны Fc и встречающийся в природе лиганд или рецептор, слитый белок Fc в которых объединены множество областей Fc, и т.п.биспецифически также включены в настоящее изобретение. Кроме того, область Fc, которая включает модификацию аминокислотного остатка, направленную на усиление или устранение эффекторной активности антитела, стабилизацию антитела и контроль периода полужизни в крови, также может быть использована для биспецифического антитела по настоящему изобретению.
В качестве биспецифического антитела или биспецифического фрагмента данного антитела по настоящему изобретению может быть использовано производное антитела, в котором присутствует радиоактивный изотоп, лекарственное средство с низкой молекулярной массой, лекарственное средство с высокой молекулярной массой, белок, лекарственное средство на основе антитела или тому подобное, включенные химически или генно-инженерным способом в биспецифическое антитело или биспецифическоий фрагмент этого антитела по настоящему изобретению.
Производное антитела по настоящему изобретению может быть получено связыванием радиоизотопа, лекарственного средства с низкой молекулярной массой, лекарственного средства с высокой молекулярной массой, иммуностимулятора, белка, лекарственного средства на основе антитела или тому подобного с N-концевой стороной или С-концевой стороной H-цепи или L-цепи биспецифического антитела или биспецифического фрагмента этого антитела по настоящему изобретению, соответствующего заместителя или боковой цепью антитела или фрагмента антитела, а также сахарной цепью или подобным из антитела или фрагмента этого антитела с использованием химического способа [Introduction to Antibody Engineering, Chijin Shokan Co., Ltd. (1994)].
Производные антител по данному изобретению могут быть также получены генно-инженерным путем: ДНК, кодирующую биспецифическое антитело или биспецифический фрагмент антитела по данному изобретению, сшивают лигазой с ДНК, кодирующей нужный белок или лекарственное антитело, и полученный полинуклеотид встраивают в экспрессионный вектор, который вводят в подходящие клетки-хозяева, и в них экспрессируется желаемый продукт.
Примеры радиоактивных изотопов включают 111In, 131I, 125I, 90Y, 64Cu, 99Tc, 77Lu, 211At и др. Радиоактивный изотоп может быть непосредственно связан с антителом, с помощью метода с использованием хлорамина Т или подобного. Также можно с антителом может быть связано вещество, хелатирующее радиоизотоп. Примеры таких хелатирующих агентов включают 1-изотиоцианатбензил-3-метилдиэтилентриаминпентауксусную кислоту (MX-DTPA) и т.п..
Примеры низкомолекулярных лекарственных веществ включают противораковые агенты, такие как алкилирующие агенты, нитрозомочевину, антиметаболиты, антибиотики, растительные алкалоиды, ингибиторы топоизомеразы, агенты гормональной терапевтии, антагонисты гормонов, ингибиторы ароматазы, ингибиторы Р-гликопротеина, комплексные соединения платины, ингибиторы фазы М или ингибиторы киназ [Clinical oncology, Japanese Journal of Cancer and Chemotherapy (1996)], противовоспалительные агенты, например стероидные соединения (гидрокортизон, преднизон и др.), нестероидные соединения, например аспирин или индометацин, иммуномодуляторы, например ауротиомалат или пеницилламин, иммуносупрессоры, например циклофосфамид или азатиоприн, антигистаминные агенты, например хлорфенирамин малеат или клемастин [Inflammation and anti-inflammatory therapy, Ishiyaku Publishers, Inc. (1982)] и проч..
Примеры противораковых агентов включают амифостин (этиол), цисплатин, дакарбазин (DTIC), дактиномицин, мехлорэтамин (мустарген), стрептозоцин, циклофосфамид, ифосфамид, кармустин (BCNU), ломустин (CCNU), доксорубицин (адриамицин), эпирубицин, гемцитабин (гемзар), даунорубицин, прокарбазин, митомицин, цитарабин, этопозид, 5-фторурацил, фторурацил, винбластин, винкристин, блеомицин, дауномицин, пепломицин, эстрамустин, паклитаксел (таксол), доцетаксел (таксотер), алдеслейкин, аспарагиназу, бусульфан, карбоплатин, оксалиплатин, недаплатин, кладрибин, камптотецин, 10-гидрокси-7-этил-камптотецин (SN38), флоксуридин, флударабин, гидроксимочевину, идарубицин, 2-меркаптоэтансульфоновую кислоту (месну), иринотекан (CPT-11), ногитекан, митоксантрон, топотекан, леупролид, мегестрол, мелфалан, меркаптопурин, гидроксикарбамид, пликамицин, митотан, пегаспаргазу, пентостатин, пипоброман, стрептозоцин, тамоксифен, госерелин, леупрорелин, флутамид, тенипозид, тестолактон, тиогуанин, тиотепу, урамустин, винорелбин, хлорамбуцил, гидрокортизон, преднизолон, метилпреднизолон, виндезин, нимустин, семустин, капецитабин, томудекс, азацитидин, тегафур/урацил (UFT), оксалоплатин, гефитиниб (иресса), иматиниб (STI571), эрлотиниб, ингибиторы FMS-подобной тирозинкиназы 3 (Flt3) ингибиторы рецептора фактора роста эндотелия сосудов (VEGFR), ингибиторы рецептора фактора роста фибробластов (FGFR), ингибиторы EGFR (например, тарцева), радицикол, 17-аллиламино-17-деметоксигелданамицин, рапамицин, амсакрин, полностью транс-ретиноевая кислота, D-пеницилламин, буцилламин, азатиоприн, мизорибин, циклоспорин, рапамицин, гидрокортизон, бексаротен (таргретин), тамоксифен, дексаметазон, прогестины, эстрогены, анастрозол (аримидекс), леуплин, аспирин, индометацин, целекоксиб, азатиоприн, пеницилламин, ауротиомалат, хлорфенирамин, малеат, хлорфенирамин, клемастин, третиноин, бексаротен, мышьяк, бортезомиб, аллопуринол, калихеамицин, ибритумомаб, тиуксетан, таргетин, озогамин, кларитромицин, лейковорин, кетоконазол, аминоглутетимид, сурамин, метотрексат или майтансиноид или их производные и т.п.
Примеры методов, применяемых для связывания низкомолекулярного лекарственного вещества с биспецифическим антителом или биспецифическим фрагментом антителапо данному изобретению, включают связывание указанного вещества с аминогруппой в молекуле антитела с использованием глутарового альдегида, связывание аминогруппы лекарственного вещества с карбоксильной группой в молекуле антитела с использованием водорастворимого карбодиимида и др.
Примеры высокомолекулярных лекарственных веществ включают полиэтиленгликоль (PEG), альбумин, декстран, полиоксиэтилен, сополимеры стирола и малеиновой кислоты, поливинилпирролидон, сополимеры пирана, гидроксипропилметакриламид и проч. В результате присоединения указанных высокомолекулярных веществ к биспецифическому антителу или фрагменту антитела по данному изобретению ожидаются такие эффекты, как (1) повышение стабильности при воздействии различных химических, физических или биологических факторов, (2) значительное продление времени полужизни в крови или (3) исчезновение иммуногенности или подавление образования антител [Bioconjugate pharmaceutical, Hirokawa-Shoten Ltd. (1993)].
Примеры методов связывания PEG с биспецифическим антителом по данному изобретению включают взаимодействие с PEGилирующими реагентамии т.п [Bioconjugate pharmaceutical, Hirokawa-Shoten Ltd. (1993)]. Примеры PEGилирующих реагентов включают модифицирующие агенты, воздействующие на ε-аминогруппу лизина (JP A-S61-178926); модифицирующие агенты, воздействующие на карбоксильную группу аспарагиновой или глутаминовой кислоты (JP- A-S56-23587); модифицирующие агенты, воздействующие на гуанидиновую группу аргинина (JP-A-H2-117920) и т.п..
Иммуностимулирующим агентом по данному изобретению может быть природный продукт, называемый иммуноадъювантом; примеры таких агентов включают вещества, усиливающие иммунную реакцию, например β(1→3)глюканы (например, лентинан или шизофиллан) или α-галактозилцерамид (KRN7000) и т.п..
Примеры белков включают цитокины или факторы роста, активирующие иммунокомпетентные клетки (например, NK-клетки, макрофаги или нейтрофилы), или токсические белки и проч.
Примеры цитокинов или факторов роста включают интерфероны (обозначаются IFN-α, IFN-β, IFN-γ), интерлейкины (обозначаются IL-2, IL-12, IL-15, IL-18, IL-21 и IL-23) колониестимулирующий фактор гранулоцитов (G-CSF), колониестимулирующий фактор гранулоцитов и макрофагов (GM-CSF), колониестимулирующий фактор макрофагов (M-CSF) и т.п.
Примеры токсических белков включают рицин, дифтерийный токсин, ONTAK и т.п., а также токсичные белки, в которые была введена мутация для контроля за токсичностью.
Антитело слитое с другим белком или лекарственным антителомможет быть получено сшиваним лигазой кДНК, кодирующую этот другой белок, с кДНК, кодирующей биспецифическое антитело или биспецифический фрагмент антитела по данному изобретению, в результате чего образуется ДНК, кодирующая слитое антитело; полученную ДНК встраивают в экспрессионный вектор, пригодный для эукариотических или прокариотических клеток, и вводят этот экспрессионный вектор в соответствующие клетки, в которых происходит экспрессия слитого антитела.
Если производное антитела по данному изобретению используют в способе выявления или количественного определения, или в качестве реагента для выявления или количественного определения, или в качестве диагностического агента, примерами вещества, которое присоединяют к биспецифическому антителу или биспецифическому фрагменту антитела по данному изобретению, включают вещества-метки, которые используют в иммунологических методах днтекции или количественного определения. Примеры веществ-меток включают ферменты, например щелочную фосфатазу, пероксидазу или люциферазу, люминесцентные вещества, например акридиновый эфир или трифенилимидазол (лофин), флуоресцентные вещества, например флуоресцеин-5-изотиоцианат (FITC) или тетраметилродамин-изотиоцианат (RITC), красители семейства Alexa (торговое название), например Alexa Fluor 488, или R-фикоэритрин (R-PE) и др.
В данное изобретение входят биспецифические антитела и биспецифические фрагменты антител, обладающие цитотоксической активностью, например активностью CDC или ADCC. Активность CDC или ADCC биспецифических антител или биспецифически фрагментов антитела по данному изобретению, направленная на клетки, в которых экспрессируется данный антиген, может быть измерена известными методами количественного определения [Cancer Immunol. Immunother., 36, 373 (1993)].
Данное изобретение относится также к композиции, содержащей в качестве активного ингредиента биспецифическое антитело или биспецифический фрагмент по данному изобретению, специфично распознающие и связывающие TfR и антиген клеточной поверхности, например EGFR, или терапевтический агент, содержащий биспецифическое антитело или биспецифический фрагмент по данному изобретению, для лечения заболевания, связанного по меньшей мере с одним из TfR и антигеном клеточной поверхности, предпочтительно такого заболевания, в каком участвуют клетки, в которых экспрессируются TfR или антиген клеточной поверхности, например EGFR.
Заболевание, связанное с TfR либо с антигеном клеточной поверхности, таким как EGFR либо с тем и другим, может быть любым, при условии, что оно связано с TfR либо с антигеном клеточной поверхности, таким как EGFR либо с тем и другим,например, злокачественная опухоль, раковое заболевание и т.п.
По данному изобретению примеры злокачественных опухолей и раковых заболеваний включают рак толстой кишки, колоректальный рак, рак легких, рак молочной железы, глиому, злокачественную меланому, рак щитовидной железы, почечноклеточную карциному, лейкозы, лимфомы, Т-клеточная лимфому, рак желудка, рак поджелудочной железы, рак шейки матки, рак тела матки, рак яичника, рак желчного протока, рак пищевода, рак печени, рак головы и шеи, рак кожи, раковые поражения мочевых путей, рак мочевого пузыря, рак предстательной железы, хориокарциному, фарингеальный рак, рак гортани, мезотелиому, опухоли плевры, арренобластому, гиперплазию эндометрия, эндометриоз, эмбриому, фибросаркому, саркому Капоши, ангиому, пещеристую гемангиому, ангиобластому, ретинобластому, астроцитому, нейрофиброму, олигодендроглиому, медуллобластому, нейробластому, глиому, рабдомиосаркому, глиобластому, остеогенную саркому, лейомиосаркому, опухоль Вильмса и проч.
В терапевтическом агенте, содержащем биспецифическое антитело или биспецифический фрагмент антитела настоящего изобретения, или их производные, могут присутствовать только биспецифическое антитело или биспецифический фрагмент антитела настоящего изобретения, или их производные, в качестве активного ингредиента, однако, в целом предпочтительно, чтобы они были в составе фармацевтического препарата, полученного одним из известных в фармацевтике методов путем из смешивания с одним или более фармацевтически приемлемым носителем.
Что касается пути введения, то предпочтителен наиболее эффективный для лечения путь введения; примеры которых включают пероральный или парентеральный, например прием внутрь через рот, введение в воздухоносные пути, интраректальное, подкожное, внутримышечное и внутривенное введение. Из них предпочтителен внутривенный путь введения.
Примеры дозированной лекарственной формы включают спреи, капсулы, таблетки, порошки, гранулы, сиропы, эмульсии, суппозитории, препараты для инъекций, мази, пластыри и др.
Доза и частота введения препаратов по данном у изобретению варьирует в зависимости от желаемого терапевтического эффекта, пути введения препарата, длительности лечения, возраста и массы тела индивида или других факторов; как правило, суточная доза для взрослых составляет от 10 мкг/кг до 10 мг/кг.
Данное изобретение относится также к реагентам для иммунологического выявления или количественного определения по меньшей мере TfR либо антигена клеточной поверхности, например, рецептора EGFR, либо обоих этих веществ; или к агентам для диагностики заболеваний, связанных по меньшей мере с TfR либо антигеном клеточной поверхности, например, EGFR, либо c обоими этими веществами, предпочтительно заболеваний, в которых участвуют клетки, где экспрессируется по меньшей мере TfR либо антиген клеточной поверхности, например, EGFR, либо оба этих вещества, причем указанные реагенты и агенты содержат биспецифические антитела или биспецифические фрагменты антител по данному изобретению. Кроме того, данное изобретение относится к способу иммунологического выявления или количественного определения по меньшей мере TfR либо антигена клеточной поверхности, например, EGFR, либо оба этих веществ; к способу лечения заболеваний, связанных по меньшей мере с TfR либо антигеном клеточной поверхности, например, EGFR, либо c обоими этими веществами, предпочтительно заболеваний, в которых участвуют клетки, где экспрессируется по меньшей мере TfR либо антиген клеточной поверхности, например, EGFR, либо оба этих вещества; или к способу диагностики заболеваний, связанных по меньшей мере с TfR либо антигеном клеточной поверхности, например, EGFR, либо c обоими этими веществами, предпочтительно заболеваний, в которых участвуют клетки, где экспрессируется по меньшей мере TfR либо антиген клеточной поверхности, например, EGFR, либо оба этих вещества; причем в указанных способах используются биспецифические антитела или биспецифические фрагменты антител по данному изобретению.
Примером способа выявления или количественного определения по меньшей мере TfR либо антигена клеточной поверхности, например, EGFR, либо обоих этих веществ, может быть какой-либо из известных в данной области техники методов. Например, это может быть иммунологический метод выявления или количественного определения.
Иммунологический метод выявления или количественного определения – это такой метод, в котором для выявления или количественного определения антитела или антигена используются меченые антиген или антитело. Примеры иммунологических методов выявления или количественного определения включают радиоиммунологический анализ (RIA), иммуноферментный анализ, в том числе твердофазный (EIA, или ELISA), флуоресцентный иммунологический анализ (FIA), люминесцентный иммунологический анализ, метод вестерн-блоттинга, физико-химические методы и др.
Путем выявления или количественного определения по меньшей мере TfR либо антигена клеточной поверхности (например, EGFR), либо обоих этих веществ с использованием биспецифических антител или биспецифических фрагментов антитела по данному изобретению возможно диагностировать заболевания, связанные по меньшей мере с TfR либо антигеном клеточной поверхности, например, EGFR, либо c обоими этими веществами, предпочтительно заболевания, в которых участвуют клетки, где экспрессируется по меньшей мере TfR либо антиген клеточной поверхности, например, EGFR, либо оба этих вещества;
Для выявления клеток, в которых экспрессируется по меньшей мере TfR либо антиген клеточной поверхности, например, EGFR, либо оба этих вещества, можно применять известные иммунологические методы, например, метод иммунопреципитации, метод иммуноцитологического окрашивания, метод имммуногистохимического окрашивания, метод окрашивания флуоресцентными антителами и другие подобные методы. Примером метода окрашивания флуоресцентными антителами является метод с использованием системы высокопроизводительного скрининга FMAT 8100 HTS (производство Applied Biosystems, Inc.) или подобные методы.
По данному изобретению биологическим образцом для выявления или количественного определения по меньшей мере TfR либо антигена клеточной поверхности,например, EGFR, либо обоих этих веществ служат клетки тканей, кровь, плазма крови, сыворотка крови, сок поджелудочной железы, моча, фекалии, тканевая жидкость, культуральная среда и др.; в выборе образца нет каких-либо ограничений, если он потенциально может содержать клетки, в которых экспрессируется по меньшей мере TfR либо антиген клеточной поверхности, например, EGFR, либо оба этих вещества.
Диагностический агент, содержащий биспецифическое антитело, или биспецифический фрагмент антитела по изобретению, или их производные, может включать реагент для осуществления взаимодействия антигена с антителом или реагент для выявления этого взаимодействия соответственно нужному методу диагностики. Примеры реагентов для осуществления взаимодействия антигена с антителом включают буферные растворы, соли и др.
Примеры реагентов для выявления взаимодействия антигена с антителом по данному изобретению включают реагенты, используемые в обычных иммунологических методах выявления или количественного определения, например меченые вторичные антитела, связывающиеся с биспецифическим антителом, или с биспецифическим фрагментом указаного антитела, или с их производным, или субстрат, соответствующий метке.
в настоящем документе конкретно описываются способ получения биспецифических антител по данному изобретению, способ определения активности биспецифических антител или биспецифических фрагментов антител, способ лечения и способ диагностики заболеваний с использованием биспецифических антител или биспецифических фрагментов антител.
1. Способ получения моноклональных антител
Способ получения моноклональных антител по данному изобретению включает следующие этапы: (1) по меньшей мере одно из очистки антигена, который должен послужить иммуногеном, и получения клеток, сверхэкспрессирующих антиген на клеточной поверхности; (2) получение клеток, продуцирующих антитела, путем иммунизации животных данным антигеном, последующий забор у них крови, определение титра антител с целью установить момент извлечения селезенки или иного материала; (3) получение миеломных клеток/миеломы; (4) слияние клеток, продуцирующих антитела, с миеломными клетками; (5) проверка полученного пула гибридом с целью выявить те, которые продуцируют искомые антитела; (6) выделение (клонирование) моноклональных клеток из пула гибридом; (7) в некоторых случаях культивирование гибридом для наработки моноклональных антител в большом количестве или размножение животных с имплантированной гибридомой; (8) изучение биологической активности полученных таким образом моноклональных антител и их антигенной специфичности или изучение свойств в качестве реагента-метки и проч.
Далее в настоящем документе подробно, по указанным выше этапам описывается способ получения моноклональных антител, связывающихся с TfR, и моноклональных антител, связывающихся с антигеном клеточной поверхности, например с EGFR, которые используются для получения биспецифических антител, связывающихся с TfR и с антигеном клеточной поверхности, например с EGFR, по данному изобретению. Способ получения антител по данному изобретению не ограничивается указанным выше: например, клетками, продуцирующими антитела, могут служить не клетки селезенки, а другие клетки, а вместо миеломных – другие бессмертные клетки.
(1) Очистка антигена
Клетки, в которых экспрессируется антиген клеточной поверхности, например EGFR, или TfR, можно получить путем введения экспрессионного вектора, содержащего кДНК, кодирующую полноразмерный антиген клеточной поверхности, например EGFR, или TfR , или его часть, в клетки E. coli, дрожжей, насекомых, животных и так далее. Также антиген клеточной поверхности, например EGFR, или TfR , или оба этих вещества можно выделить из культур различных опухолевых клеток или тканей человека, где интенсивно экспрессируется антиген клеточной поверхности, например EGFR, или TfR , или оба этих вещества, очистить и использовать как антиген. Также антигеном могут служить сами культивируемые опухолевые клетки или ткани. Кроме того, синтетический пептид с аминокислотной последовательностью части молекулы антигена клеточной поверхности, например EGFR, или TfR, можно получить путем химического синтеза (с использованием Fmoc или tBoc), и также использовать его в качестве антигена.
Антиген клеточной поверхности, например EGFR, или TfR , используемые по данному изобретению, могут быть получены способами, описанными в руководствах Molecular Cloning, A Laboratory Manual, 2-е издание, Cold Spring Harbor Laboratory Press (1989), или Current Protocols In Molecular Biology, John Wiley & Sons (1987-1997), или подобных им, например, путем экспрессии ДНК, кодирующей антиген клеточной поверхности, например EGFR, или TfR , в клетках-хозяевах следующим образом.
В подходящий экспрессионный вектор ниже промотора встраивают полноразмерную кДНК, содержащую область, кодирующую антиген клеточной поверхности, например EGFR, или TfR . Вместо полноразмерной кДНК можно использовать фрагмент подходящей длины, полученный из полноразмерной кДНК и содержащий область, кодирующую нужный полипептид. Полученный рекомбинантный вектор вводят в клетки-хозяева, пригодные для его экспрессии, и трансформированные клетки продуцируют антиген клеточной поверхности, например EGFR, или TfR .
В качестве экспрессионного вектора можно использовать любой вектор, коль скоро он способен автономно реплицироваться в выбранных клетках-хозяевах или же может встраиваться в хромосомную ДНК и содержит подходящий промотор в положении, пригодном для транскрипции ДНК, кодирующей антиген клеточной поверхности, например EGFR, или TfR .
В качестве клеток-хозяев можно брать любые клетки, например микроорганизмы, принадлежащий к роду Escherichia, например E. coli, дрожжи, клетки насекомых или клетки животных, , или иные клетки, при условии, что в них может экспрессироваться нужный ген.
При использовании в качестве клеток-хозяев клеток прокариот, например E. coli, предпочтителен рекомбинантный вектор, способный автономно реплицироваться в прокариотических клетках, а также включающий промотор, сайт связывания с рибосомой, ДНК, содержащую область, кодирующую антиген клеточной поверхности, например EGFR, или TfR , и последовательность терминации транскрипции. Терминатор транскрипции не обязателен в рекомбинантном векторе, но, если он имеется, то предпочтительно, чтобы терминирующая последовательность располагалась сразу после структурного гена. Также рекомбинантный вектор может содержать ген, контролирующий промотор.
В качестве рекомбинантного вектора предпочтительна плазмида, в которой должным образом подобрано расстояние между последовательностью Шайна-Дальгарно (сайт связывания рибосомы) и стартовым кодоном (например, оно составляет 6-18 нуклеотидов).
В последовательности ДНК, кодирующей антиген клеточной поверхности, например EGFR, или TfR , можно заменить некоторые нуклеотиды, чтобы кодоны стали оптимальными для экспрессии в данном виде клеток-хозяев и таким образом увеличилась продукция этими клетками антигена клеточной поверхности, например EGFR, или TfR,
В качестве экспрессионного вектора может служить любой вектор, при условии, что он способен выполнять нужную функцию в выбранных клетках-хозяевах; примеры таких векторов включают pBTrp2, pBTac1, pBTac2 (производство Roche Diagnostics K.K.), pKK233-2 (производство Pharmacia Corporation), pSE280 (производство Invitrogen, Inc.), pGEMEX-1 (производство Promega Corporation), pQE-8 (производство QIAGEN, Inc.), pKYP10 (JP- A-S58-110600), pKYP200 [Agricultural Biological Chemistry, 48, 669 (1984)], pLSA1 [Agric. Biol. Chem., 53, 277 (1989)], pGEL1 [Proc. Natl. Acad. Sci. USA, 82, 4306 (1985)], pBluescript II SK(-) (производство Stratagene Corporation), pTrs30 [получен из E. coli JM109/pTrS30 (FERM BP-5407)], pTrs32 [получен из E. coli JM109/pTrS32 (FERM BP-5408)], pGHA2 [получен из E. coli IGHA2 (FERM BP-400), JP- A-S60-221091], pGKA2 [получен из E. coli IGKA2 (FERM BP-6798), JP- A-S60-221091], pTerm2 (патенты США №№ 4,686,191, 4,939,094 или 5,160,735), pSupex, pUB110, pTP5, pC194, pEG400 [J. Bacteriol., 172, 2392 (1990)], pGEX (производство Pharmacia Corporation), pET System (производство Novagen, Inc.), pME18SFL3 (производство Toyobo Co., Ltd.) и т.п..
В качестве промотора можно использовать любой промотор, способный функционировать в выбранных клетках-хозяевах. Примеры таких промоторов включают промоторы из E. coli, промоторы из бактериофагов или подобные им, например промотор триптофанового (trp) оперона (Ptrp), промотор лактозного (lac) оперона, a также промоторы PL и PR бактериофага λ или промотор бактериофага T7. Кроме того, примеры промоторов, используемых по данному изобретению, включают искусственно сконструированные и модифицированные промоторы, например тандемный промотор, в котором два промотора Ptrp соединены друг за другом; промоторы tac, lac-T7 или let I и т.п.
Примеры клеток-хозяев, используемых по данному изобретению, включают E. coli XL1-Blue, E. coli XL2-Blue, E. coli DH1, E. coli MC1000, E. coli KY3276, E. coli W1485, E. coli JM109, E. coli HB101, E. coli No. 49, E. coli W3110, E. coli NY49, E. coli DH5α, и т.п.
Введение рекомбинантного вектора в клетки-хозяева можно осуществлять любым методом, применяемым для введения ДНК в клетки; примеры таких методов включают методы с использованием ионов кальция [Proc. Natl. Acad. Sci. USA, 69, 2110 (1972), Gene, 17, 107 (1982), Molecular & General Genetics, 168, 111 (1979)].
В случае использования животных клеток в качестве клеток-хозяев для введения экспрессионного вектора, таким вектором может быть любой пригодный для клеток животных, например pcDNAI (производство Invitrogen, Inc.), pcDM8 (производство Funakoshi Co., Ltd.), pAGE107 [JP- A-H3-22979; Cytotechnology, 3, 133 (1990)], pAS3-3 (JP- A-H2-227075), pCDM8 [Nature, 329, 840 (1987)], pcDNAI/Amp (производство Invitrogen, Inc.), pcDNA3.1 (производство Invitrogen, Inc.), pREP4 (производство Invitrogen, Inc.), pAGE103 [J. Biochemistry, 101, 1307 (1987)], pAGE210, pME18SFL3, pKANTEX93 (WO 97/10354) и т.п.
Промотором по данному изобретению может служить любой промотор, способный функционировать в животных клетках; примеры таких промоторов включают промотор немедленно ранних (IE) генов цитомегаловируса (CMV), промотор ранних генов вируса SV40, ретровирусный промотор, промоторы генов металлотионеинов, промоторы генов белков теплового шока, промотор SRα, промотор или энхансер вируса лейкоза мышей Молони. Также можно использовать вместе с промотором энхансер немедленно ранних генов цитомегаловируса человека.
Примеры клеток-хозяев по данному изобретению включают человеческие клетки лимфомы Беркитта линии Namalwa, почечные клетки COS африканской зеленой мартышки, клетки яичника китайского хомячка СНО, человеческие лейкозные клетки HBT5637 (JP- A-S63-000299) и др.
Введение рекомбинантного вектора в клетки-хозяева можно осуществлять любым методом, применяемым для введения ДНК в клетки; примеры таких методов включают электропорацию [Cytotechnology, 3, 133 (1990)], метод с использованием фосфата кальция (JP- A-H2-227075), липофекцию [Proc. Natl. Acad. Sci. USA, 84, 7413 (1987)] и др.
Антиген клеточной поверхности, например EGFR или TfR , можно получать, культивируя микроорганизмы, в клетках которых имеется рекомбинантный вектор, включающий ДНК, кодирующую антиген клеточной поверхности, например EGFR или TfR , или трансформанты полученныеих животных клеток, или иные клетки, полученные, как описано выше: в соответствующих культуральной среде и условиях, обеспечивающих продукцию TfR , или антигена клеточной поверхности, например EGFR, или оба этих вещества, микроорганизмом или трансформантом и их накопление в культуральной среде, откуда их выделяют. Культивирование трансформантов осуществляется обычными методами, используемыми для культивирования клеток-хозяев
В тех случаях, когда экспрессия TfR или антигена клеточной поверхности, например EGFR, осуществляется в клетках эукариотического происхождения, можно получать TfR или антиген клеточной поверхности, например EGFR с присоединенными к ним остатками сахаров или сахарных цепейк.
При культивировании микроорганизмов, трансформированных рекомбинантным вектором с индуцибельным промотором, в культуральную среду при необходимости добавляют индуктор. Например, в случае культивирования микроорганизмов, трансформированных рекомбинантным вектором с lac-промотором, в культуральную среду добавляют изопропил-β-D-тиогалактопиранозид или подобное соединение, а в случае культивирования микроорганизмов, трансформированных рекомбинантным вектором с trp-промотором, в культуральную среду добавляют индолакриловую кислоту или подобное соединение.
Примеры культуральных сред, пригодных для культивирования трансформантов, полученных из животных клеток, включают такие обычно используемые среды, как среда RPMI 1640 [The Journal of the American Medical Association, 199, 519 (1967)], среда Игла MEM [Science, 122, 501 (1952)], среда Игла MEM, модифицированная по Дульбекко [Virology, 8, 396 (1959)], среда 199 [Proc. Soc. Exp. Biol. Med., 73, 1 (1950)], среда Дульбекко, модифицированная по Искову (IMDM), или указанные среды с добавлением эмбриональной бычьей сыворотки (FBS) или подобного ингредиента, или другие подобные среды. Культивирование обычно проходит в течение 1-7 суток при температуре 30-40°C в атмосфере, содержащей 5% CO2; рН культуральной среды 6-8. При необходимости в процессе культивирования в культуральную среду добавляют антибиотик, например канамицин или пенициллин.
Для экспрессии генов, кодирующих такие антигены клеточной поверхности, EGFR или TfR , помимо прямой экспрессии можно применять метод продуцирования с секрецией, экспрессию слитого белка и др. [Molecular Cloning, A Laboratory Manual, 2-е издание, Cold Spring Harbor Laboratory Press (1989)]. Примеры методов получения таких антигенов клеточной поверхности, как EGFR или TfR , включают продуцирование в клетках-хозяевах, секретирование указанных продуктов из клеток-хозяев и продуцирование на внешней мембране клеток-хозяев; наиболее подходящий метод подбирают, изменяя клетки-хозяева или структуру антигена клеточной поверхности, такого как EGFR или TfR, который должен продуцироваться.
Например, можно осуществить продуцирование белка, слитого с антигеном, получив ДНК, в которой ДНК, кодирующая область Fc антитела, ДНК, кодирующая глутатион-S-трансферазу (GST), ДНК, кодирующая метку FLAG или ДНК, кодирующая Гистидиновую метку, или иная нуклеотидная последовательность лигирована с ДНК, кодирующей аминокислотную последовательность внеклеточного домена, с последующей экспрессией и очисткой. Конкретные примеры такого подхода включают получение Fс-слитого белка, в котором внеклеточный домен антигена клеточной поверхности (EGFR или TfR) соединен с областью Fc человеческого IgG, и слитый белок, в котором внеклеточный домен антигена клеточной поверхности, такой как EGFR или TfR, соединен с глутатион-S-трансферазой (GST).
При продуцировании антигена клеточной поверхности, например EGFR или TfR в клетке-хозяине или на внешней мембране клетки-хозяина, поверхностные антигены типа EGFR или TfR можно активно секретировать во внеклеточное пространство, применяя методы, описанные в работах Рaulson et al. J. Biol. Chem., 264, 17619 (1989), Lowe et al. Proc. Natl. Acad. Sci., USA, 86, 8227 (1989), Genes Develop., 4, 1288 (1990) или в JP- A-H05-336963, в публикации WO 94/23021 или подобные методы. Кроме того, количество продуцируемого антигена клеточной поверхности, например EGFR или TfR, можно увеличить с помощью системы амплификации нужных генов с использованием гена дигидрофолатредуктазы или подобные методы (см. JP- A-H2-227075).
Продуцируемый клетками-хозяевами антиген клеточной поверхности (например, EGFR или TfR ) выделяют и очищают, например, следующим образом.
Если антиген клеточной поверхности, например EGFR или TfR , экспрессировавшийся в клетке-хозяине, оказывается в растворенном состоянии, то по окончании культивирования клеток-хозяев их собирают путем центрифугирования, суспендируют в водном буферном растворе, гомогенизируют с помощью ультразвукового гомогенизатора, френч-пресса, гомогенизатора Мантона-Гаулина (компания Manton Gaulin Manufacturing Co., Inc.), бисерной мельницы Dyno-Mill или других подобных устройств; в результате получается раствор бесклеточного экстракта. Этот раствор центрифугируют и из полученного супернатанта выделяют нужный белок и очищают его, применяя обычные методы выделения и очистки белков, например экстракцию растворителем, высаливание сульфатом аммония или другой солью, обессоливание, осаждение с использованием органических растворителей, анионобменную хроматографию на таких смолах, как диэтиламиноэтил (DEAE)-сефароза или DIAION HPA-75 (производство Mitsubishi Chemical Corporation), катионобменную хроматографию на таких смолах, как S-сефароза FF (производство Pharmacia Corporation), гидрофобную хроматографию на таких смолах, как бутилсефароза или фенилсефароза, гель-фильтрацию на молекулярных ситах, аффинную хроматографию, хроматофокусирование, электрофорез, включая изоэлектрофокусирование и другие методы по отдельности или в сочетаниях.
Если экспрессирующийся в клетке-хозяине антиген клеточной поверхности, например EGFR или TfR , образует нерастворимые частицы, поступают следующим образом. Клетки собирают и гомогенизируют, как описано выше, и затем центрифугируют, так что нерастворимые частицы антигена клеточной поверхности, например EGFR или TfR , оказываются в осадке. Собранные нерастворимые частицы антигена клеточной поверхности, например, EGFR или TfR, солюбилизируют с помощью агента, денатурирующего белки. Нормальную конформацию антигена клеточной поверхности (EGFR или TfR ) восстанавливают путем разбавления или диализа солюбилизованной фракции. После этого антиген клеточной поверхности выделяют и очищают указанными выше методами.
Если антиген клеточной поверхности, например EGFR или TfR, или его производное, например модифицированная сахарами форма, секретируется клеткой-хозяином во внеклеточное пространство, то антиген клеточной поверхности, например EGFR или TfR, или их производное, например модифицированная сахарами форма, может быть собрана из супернатанта культуральной среды. Тогдасупернатант культуральной среды подвергают обработке описанными выше методами, включая центрифугирование, и в результате получают растворимую фракцию, из которой выделяют и очищают описанными выше методами искомый белок.
Также антиген клеточной поверхности, например EGFR или TfR , используемый по данному изобретению, можно получить путем химического синтеза с использованием методов Fmoc или tBoc. Говоря конкретно, химический синтез осуществляют с помощью пептидного синтезатора производства Advanced Chemtech, Inc., PerkinElmer, Inc., Pharmacia Corporation, Protein Technology Instrument, Inc., Synthecell-Vega Biomolecules Corporation, Perceptive, Inc., Shimadzu Corporation или других подобных компаний.
(2) Получение клеток, продуцирующих антитела.
Полученным согласно (1) антигеном иммунизируют животных, например мышей, крыс, хомяков, кроликов, крупный рогатый скот или альпаку, и из селезенки, лимфатических узлов или периферической крови иммунизированного животного выделяют клетки, продуцирующие антитела. Животными для иммунизации нужным антигеном могут служить также, например. трансгенные мыши, у которых образуются антитела человеческого происхождения, описанные в работе Tomizuka. et al., Proc. Natl. Acad. Sci. USA., 97, 722 (2000); мыши с условным нокаутом гена, кодирующего TfR или антиген клеточной поверхности, например, EGFR, для усиления иммуногенности.
Иммунизация животных осуществляется путем введения антигена вместе с подходящим адъювантом, например полным адъювантом Фрейнда, гелем гидроксида алюминия, вакциной Bordetella pertussis и др. Введение иммуногена мышам может осуществляться любым способом, например путем подкожной инъекции, внутривенной инъекции, внутрибрюшинной инъекции, интрадермальной инъекции, внутримышечной инъекции, инъекции в подушечку лапы и др; предпочтительны внутрибрюшинная и внутривенная инъекции и инъекция в подушечку лапы. В тех случаях, когда антиген представляет собой частичный пептид, получают конъюгат этого антигена с белком-носителем, например с бычьим сывороточным альбумином (BSA) или гемоцианином Fissurella aperturа (KLH) и используют этот конъюгат как иммуноген.
Антиген вводят животным 5-10 раз с интервалом 1-2 недели. Спустя 3-7 суток после каждого введения у иммунизированного животного берут кровь из ретроорбитального или подъязычного венозного сплетения и в сыворотке взятой крови определяют титр антител методом иммуноферментного анализа [Antibodies - A Laboratory Manual, Cold Spring Harbor Laboratory (1988)] или иным методом. Если источником клеток, продуцирующих антитела, которые затем используются при слиянии служат животные, у которых титр антител к антигену, использовавшемуся для иммунизации, в сыворотке крови оказался достаточным, то эффект от последующей обработки усиливается.
Через 3-7 суток после последнего введения антигена у иммунизированных животных извлекают ткани, содержащие клетки, продуцирующие антитела, например селезенку, и выделяют эти клетки. Клетками, продуцирующими антитела, являются лимфоциты – плазматические клетки и их предшественники. Эти клетки можно получить из любого участка организма, но, как правило, используют селезенку, костный мозг, миндалины, периферическую кровь или сочетания указанных источников; чаще всего используются клетки селезенки. Для выделения клеток, продуцирующих антитела, из селезенки, ее ткань измельчают, делают суспензию, которую центрифугируют, удаляют эритроциты, а полученные клетки, продуцирующие антитела, суспендируют в подходящей среде.
(3) Получение миеломы
Для получения миеломы можно использовать клетки млекопитающих, например мышей, крыс, морских свинок, хомяков, кроликов или человека, не способные самостоятельно продуцировать антитела; как правило, используют стандартные клеточные линии мышиных клеток, например происходящие из мышей BALB/c резистентные к 8-азагуанину миеломные клетки линий P3-X63Ag8-U1 (P3-U1) [Current Topics in Microbiology and Immunology, 18, 1 (1978)], P3-NS1/1-Ag41 (NS-1) [European J. Immunology, 6, 511 (1976)], SP2/0-Ag14 (SP-2) [Nature, 276, 269 (1978)], P3-X63-Ag8653 (653) [J. Immunology, 123, 1548 (1979)], P3-X63-Ag8 (X63) [Nature, 256, 495 (1975)] или им подобные. Выращивают субкультуру таких клеток в подходящей среде, например в среде с 8-азагуанином [cреда RPM1640, содержащая глутамин, 2-меркаптоэтанол, гентамицин, эмбриональную телячью сыворотку (FCS) и 8-азагуанин], среде Дульбекко, модифицированной по Искову (IMDM), среде Игла, модифицированной по Дульбекко (DMEM), и др. Указанные выше клеточные линии субкультивируют в нормальной среде (например, в среде DMEM, содержащей 10% FCS) в течение 3-4 суток перед слиянием, чтобы в день осуществления слияния имелось 2 x 107 или более клеток.
(4) Слияние клеток
Клетки, продуцирующие антитела (APC), полученные в (2), и миеломные клетки (MC), полученные в (3), хорошо промывали минимальной основной средой (MEM) или PBS (1.83 г двузамещенного фосфата натрия; 0,21 г однозамещенного фосфата калия; 7,65 г хлорида натрия; 1 л дистиллированной воды; рН 7,2) и смешивали для получения соотношения Клетки, продуцирующие антитела: миеломные клетки от 5:1 до 10:1, затем центрифугировали и супернатант отбрасывали. Осадок клеток тщательно разрыхляли и добавляли смешанный раствор полиэтиленгликоля 1000 (PEG-1000), среды MEM и диметилсульфоксида при перемешивании и температуре 37°C. Затем прибавляли среду МЕМ: сначала несколько раз с интервалом 1-2 минуты по 1-2 мл, затем до суммарного объема 50 мл. Центрифугировали, супернатант отбрасывали, осадок клеток осторожно разрыхляли и суспендировали в среде HAT (нормальная культуральная среда, содержащая гипоксантин, тимидин и аминоптерин. Полученную суспензию культивировали в инкубаторе с атмосферой, содержащей 5% CO2, при температуре 37°C в течение 7-14 суток.
Кроме того, слияние клеток можно проводить следующим образом. Клетки селезенки и миеломные клетки хорошо промывают бессывороточной средой (например, DMEM) или солевым раствором, забуференным фосфатом [далее в настоящем документе называемым фосфатно-солевым буферным раствором ] и смешивают в соотношении клетки селезенки: миеломные клетки около 5:1 - 10:1, затем центрифугируют. Супернатант удаляют, осадок клеток тщательно разрыхляют и добавляют по каплям при перемешивании 1 мл бессывороточной среды, содержащей 50% (масса/объем) полиэтиленггликоля с молекулярной массой 1000-4000. Затем медленно прибавляют 10 мл бессывороточной среды, после чего центрифугируют. Супернатант удаляют, осевшие клетки суспендируют в нормальной культуральной среде, содержащей необходимое количество раствора гипоксантина-аминоптерина-тимидина (НАТ) и человеческий интерлейкин-2 ( IL-2) (далее в настоящем документе эта среда обозначается HAT), Полученную суспензию вносят в лунки культурального планшета (далее называется просто планшетом) и культивируют в атмосфере, содержащей 5% углекислого газа при температуре 37°C в течение 2 недель. В процессе культивирования прибавляют по необходимости среду НАТ.
(5) Отбор гибридом
Если для слияния используются миеломные клетки линии с резистентностью к 8-азагуанину, то есть клетки с дефицитом гипоксантин-гуанин-фосфорибозилтрансферазы (HGPRT), то не слившиеся миеломные клетки и миеломные клетки, слившиеся друг с другом, не выживают в среде HAТ. С другой стороны продукты слияния клеток, продуцирующих антитела, друг с другом, и гибридомы – продукты слияния клеток, продуцирующих антитела, с миеломными клетками – могут жить в среде НАТ, однако продолжительность жизни слившихся друг с другом клеток, продуцирующих антитела, существенно короче. Таким образом, при длительном культивировании в среде НАТ выживают только гибридомы – продукты слияния клеток, продуцирующих антитела, с миеломными клетками, и тем самым достигается отбор гибридом.
Чтобы из отдельных гибридом образовались колонии, заменяют культуральную среду: из среды НАТ исключают аминоптерин (далее такая среда обозначается НТ). Из каждой полученной культуры отбирают порцию супернатанта и можно выбрать продуцирующую антитело гибридому используя упомянутый ниже способ измерения титра антител. Таким образом выявляют гибридомы, продуцирующие антитела. Примеры методов количественного определения титра антител включают различные известные методы, такие как радиоиммунологический анализ (метод RIA), твердофазный иммуноферментный анализ (метод ELISA), метод флуоресцентного мечения антител и реакцию пассивной гемагглютинации; из них предпочтительны RIA или ELISA в силу большей чувствительности, быстроты выполнения, точности, возможности автоматизации и др.
Те гибридомы, которые по результатам определения титра антител продуцируют нужные антитела, переносят в другой планшет и клонируют. Примеры методов клонирования включают метод предельных разведений, где культуру разбавляют так, чтобы в одной лунке планшета содержалась одна клетка, метод культивирования на мягком агаре, где култивирование проводится в мягком агаре для сбора колоний, метод, в котором отдельные клетки отбирают с использованием микроманипулятора, метод, в котором отдельные клетки отбирают с использованием клеточного сортера, , и др.
В случае лунок, в которых наблюдался титр антител, клонирование повторяют 2-4 раза, применяя, например, метод предельных разведений; отбирают те клетки, для которых неизменно наблюдается достаточный титр антител, и используют их как гибридомный штамм, продуцирующий моноклональные антитела к TfR или антигену клеточной поверхности, например EGFR.
(6) Получение моноклональных антител
Моноклональные гибридомы, продуцирующие антитела, полученные в (5) вводят путем внутрибрюшинной инъекции мышам или голым мышам возрастом 8-10 недель, которым до этого ввели 0,5 мл 2,6,10,14-тетраметилпентадекана (пристан) и затем содержат в течение 2 недель. Через 10-21 сутки гибридома вызывает образование асцитной опухоли. У мышей извлекают эти опухоли, твердое вещество отделяют путем центрифугирования, оставшуюся жидкость высаливают 40-50%-ным сульфатом аммония и осажденное вещество очищают путем осаждения каприловой кислотой, на колонке с DEAE-сефарозой или с протеином A или путем гель-фильтрации; собирают фракции IgG или IgM. Таким образом получаются очищенные моноклональные антитела. Также можно растить гибридому в брюшной полости мыши, например линни BALB/c) или гомозиготной голой мыши (Nu/Nu), крысы, хомяка, морской свинки, кролика или другого животного и там в асците будут в большом количестве образовываться моноклональные антитела, связывающиеся с TfR или с антигеном клеточной поверхности, например EGFR.
После культивтрования полученной в (5) гибридомы, продуцирующей моноклональные антитела, в среде RPMI 1640, содержащей 10% FBS, или в другой подобной среде; супернатант отделяют путем центрифугирования, осадок суспендируют в среде GIT, среде SFM для гибридом, содержащей 5% GF21 (фирмы Daigo) или в подобной среде. Полученную суспензию культивируют в течение 3-7 суток в колбе, роллерным методом, в культуральном пакете или иным способом. Полученную суспензию клеток центрифугируют, супернатант пропускают через колонку с протеином А или G и собирают фракцию IgG. Таким образом получают очищенные моноклональные антитела. Также возможен простой способ очистки с использованием имеющихся в продаже наборов для очистки моноклональных антител, например MabTrap GII kit производства Amersham Pharmacia Biotech, Inc. и т.п.
Подкласс антител определяют путем иммуноферментного анализа с помощью наборов для типирования. Количественное определение белка делают по методу Лоури или рассчитывают по поглощению на длине волны 280 нм [ 1,4(OD280) = 1 мг иммуноглобулина/мл].
(7) Анализ связывания моноклонального антитела
с TfR или с антигеном клеточной поверхности, таким как (EGFR)
Активность связывания моноклонального антитела с TfR или с антигеном клеточной поверхности, например EGFR, может быть измерена в системе анализа связывания, такой как способ по Оухтерлони, ELISA, RIA, проточная цитометрия (FCM) или метод поверхностного плазмонного резонанса (SPR).
Метод Оухтерлони прост в выполнении, но при низкой концентрации антител приходится проводить концентрирование. С другой стороны если использовать метод ELISA или RIA, то при условии непосредственного взаимодействия полученного из культуры клеток супернатант с адсорбированным на твердой фазе антигеном и последующего использования вторичных антител, соответствующих различным изотипам и подклассам иммуноглобулинов, возможно идентифицировать изотип и подкласс нужного антитела и количественно определить его активность связывания.
В качестве конкретного примера анализа: очищенный или частично очищенный рекомбинантный антиген клеточной поверхности, например EGFR или TfR , адсорбируют на твердой фазе в 96-луночном планшете для ELISA, затем ту поверхность твердой фазы, где нет адсорбированного антигена, блокируют при помощи белка, не родственного данному антигену, например бычьего сывороточного альбумина (BSA). После ELISA планшет промывают солевым раствором, забуференным фосфатом (PBS) и PBS, содержащим 0,05% полисорбата-20 (Tween 20) (Tween-PBS), проводят реакции с серийными разведениями первого антитела (например, мышиной сыворотки крови, супернатанта культуры клеток и др.), и эти антитела, связываются с антигеном, иммобилизованным на твердой фазе Затем в лунки планшета добавляют вторичные антитела – антииммуноглобулиновые антитела, меченные биотином, ферментом (например, пероксидазой хрена (HRP), щелочной фосфатазой (ALP) или другим), хемилюминесцентным агентом, радиоактивным агентом или иным образом, и эти вторичные антитела связываются с первыми. После тщательного промывания лунок Tween-PBS, проводят выявляющую реакцию соответственно взятой метке вторичного антитела и таким образом отбирают моноклональные антитела, специфично взаимодействующие с антигеном-мишенью.
Метод FCM позволяет измерить активность связывания данного антитела с клетками, в которых экспрессируется соответствующий антиген [Cancer Immunol. Immunother. 36, 373 (1993)]. Связывание антитела с антигенным мембранным белком на клеточной мембране означает, что данное антитело распознает конформацию природного антигена и связывается с ним.
Примеры применения метода SPR включают кинетический анализ посредством Biacore. Например, используя Biacore T100 можно измерить кинетику связывания антигена с тестируемым веществом и проанализировать полученные данные с помощью программного обеспечения этого прибора. В качестве конкретного примера процедуры После фиксирования антитела к мышиному IgG на чипе CM5 путем иммобилизации по аминогруппе; вводят поток тестируемой субстанции, например супернатанта культуры гибридом или очищенного моноклонального антитела, чтобы она связаласьв соотвествующем количестве; затем вводят поток антигена в различных известных концентрациях и измерили связывание и диссоциацию. Проводят кинетический анализ полученных данных, исходя из связывания 1:1, с помощью программного обеспечения прибора и определяют различные кинетические параметры. Альтернативно, на сенсорном чипе, например путем иммобилизации по аминогруппе, фиксируют антиген клеточной поверхности, например EGFR или TfR , вводят поток раствора очищенного моноклонального антитела при множестве известных концентраций и измеряют связывание и диссоциацию. Проводят кинетический анализ полученных данных, исходя из бивалентного связывания, с помощью программного обеспечения прибора и определяют различные параметры.
Также по данному изобретению можно отбирать антитела, связывающиеся с TfR или с антигеном клеточной поверхности, например с EGFR, конкурентно с антителом против TfR или антигена клеточной поверхности, например EGFR, если проводить анализ в Biacore таким образом, что аналиты присутствуют в описанной выше системе для связывания одновременно. Таким образов проводя скриниг антител, чье связывание с данным антигеном подавляется при добавлении тестируемого антитела, можно получить антитела, конкурирующие с полученным выше антителом за связывание с TfR или с антигеном клеточной поверхности, например с EGFR.
(8) Идентификация эпитопа моноклонального антитела
к TfR или антигену клеточной поверхности, например ЕGFR
В настоящем изобретении эпитоп, который распознает и связывает антитело по данному изобретению, идентифицируют следующим образом.
Например, получают частично дефектный вариант антигена. или мутантный вариант антигена, в котором модифицирован аминокислотный остаток, различающийся у разных видов, или мутантный вариант антигена, в котором модифицирован определенный домен, и если реактивность антитела в отношении дефектного или мутантного варианта снижена, то, значит, этот дефектный сайт или сайт, где была модифицированана аминокислота, представляет собой эпитоп, к которому специфично данное антитело. Такой частично дефектный вариант антигена или мутантный антиген может быть получен как секреторный белок при помощи подходящих клеток-хозяева, например E. coli, дрожжей, растительных клеток, или клеток млекопитающих и подобных,; или такой антиген может быть получен в формате антиген-экспнрессирующей клетки, , путем его экспрессии на мембране подходящих клеток-хозяев. В случае антигена, связанного с клеточной мембраной, нужно, чтобы при его экспрессии получалась должная конформация антигенного белка, поэтому предпочтительна экспрессия на мембране клетки-хозяина. Также для определения реакционной способности антитела в отношении данного антигена можно получить синтетический пептид, имитирующий первичную структуру или конформацию антигена. Например, известными методами пептидного синтеза получают различные участки аминокислотной последовательности антигена.
Например, в случае внеклеточного домена TfR или антигена клеточной поверхности, такого как EGFR, человека или мыши, можно идентифицировать эпитоп, к которому специфично данное антитело, путем создания химерного белка, в котором определенным образом скомбинированы домены, представляющие соответствующие участки, и проверки реакционной способности данного антитела в отношении этого белка. Можно также более детально определить эпитоп, к которому специфично данное антитело, синтезируя различные олигопептиды соответствующих участков или получая мутантные варианты нужного пептида с помощью методов олигопептидного синтеза, известных специалистам в данной области техники, и затем проверяя реакционную способность данного антитела в отношении этих пептидов. Простой способ получения различных олигопептидов - использование имеющихся в продаже специальных наборов [например, SPOTs Kit производства Genosys Biotechnologies, Inc.; линия наборов для множественного параллельного синтеза пептидов (технология Multipin) производства Chiron Corporation и др.], в которых применяется технология Multipin или подобный метод.
Антитело, связывающееся с тем же эпитопом, что и антитело, связывающееся с TfR или с антигеном клеточной поверхности, например EGFR; может быть получено путем идентификации эпитопа полученного антитела при помощи вышеописанной системы анализа связывания, в затем получают сиететический пептид, являющийся частью этого эпитопа, синтетический пептид, имитирующий конформацию данного эпитопа, или рекомбинантный эпитоп, или иной его вариант и полученным пептидом проводят иммунизацию.
Например, если эпитоп принадлежит мембранному белку, то антитело, специфичное к этому эпитопу, можно более эффективно получать путем создания рекомбинантного слитого белка, в котором весь внеклеточный домен целиком или его часть присоединены к соответствующей метке, например к тэгу FLAG или к гистидиновому тэгу, или к белку GST, или к области Fc антитела и др., и этим рекомбинантным белком проводят иммунизацию.
2. Получение генно-инженерных рекомбинантных антител
Примеры получения генно-инженерных рекомбинантных антител в общих чертах описаны в работах P. J. Delves., Antibody Production Essential Techniques., 1997 Wiley; P. Shepherd, C. Dean. Monoclonal Antibodies., 2000 Oxford University Press; J. W. Goding., Monoclonal Antibodies: principles and practice., 1993 Academic Press, и в других подобных источниках; тем не менее, методы получения химерных антител, гуманизированных антител и человеческих антител описываются ниже в настоящем документе. Также этими же методами могут быть получены генно-инженерные рекомбинантные антитела мышей, крыс, хомяков и кроликов.
(1) Получение кДНК, кодирующей V область моноклонального антитела, продуцируемого гибридомой
Комплементарную ДНК (кДНК),, кодирующую кодирующую VH и VL моноклонального антитела получают, например, следующим образом.
Вначале из гибридомы, продуцирующей моноклональное антитело, выделяют мРНК и синтезируют по ним кДНК. Затем синтезированные кДНК клонируют по отдельности в векторе, например в фаге или плазмиде, получая таким образом библиотеку кДНК. Из этой библиотеки выделяют рекомбинантный фаг или рекомбинантную плазмиду, содержащую кДНК, кодирующую VH или VL, используя в качестве зонда ДНК, кодирующую константную или вариабельную область антитела соответственно. Определяют полную нуклеотидную последовательность, кодирующую VH или VL в выделенном рекомбинантном фаге или в рекомбинантной плазмиде, а затем по нуклеотидной последовательности выводят полную аминокислотную последовательность VH или VL.
Для получения гибридом используют обычно используеют животных, не являющихся человеком, например мышей, крыс, хомяков, кроликов или т.п., но можно использовать любое животное, если можно в результате получить гибридому.
Для получения тотальной РНК из гибридомы применяют метод с использованием гуанидинтиоцианата и трифторацетата цезия [Methods in Enzymol., 154, 3 (1987)] или готовый набор, например RNA easy Kit (производство QIAGEN, Inc.) или т.п.
Матричную РНК выделяют из тотальной РНК на колонках с олиго(dT)-целлюлозой [Molecular Cloning, A Laboratory Manual, 2-е издание, Cold Spring Harbor Laboratory Press (1989)] или с помощью готовых наборов, например Oligo-dT30 <Super> mRNA Purification Kit (производство Takara Bio, Inc.) и т.п. Для получения мРНК можно также использовать такие наборы, как Fast Track mRNA Isolation Kit (производство Invitrogen, Inc.) или QuickPrep mRNA Purification Kit (производство Pharmacia Corporation).
Для синтеза кДНК и получения библиотеки кДНК применяют известный метод, описанный, например, в руководствах Molecular Cloning, A Laboratory Manual, 2-е издание, Cold Spring Harbor Laboratory Press (1989), Current Protocols in Molecular Biology, Supplement 1, John Wiley & Sons (1987-1997), или используют готовые наборы, например SuperScript Plasmid System for cDNA Synthesis and Plasmid Cloning, (производство Invitrogen, Inc.) или ZAP-cDNA Synthesis Kit (производство Stratagene Corporation) и т.п..
Когда получена библиотека кДНК, в качестве вектора, в который встраивают кДНК, синтезированную по мРНК, которая выделена из гибридомы, можно использовать любой вектор, в который эту кДНК возможно встроить.
Например, пригодны такие векторы, как ZAP Express [Strategies, 5, 58 (1992)], pBluescript II SK(+) [Nucleic Acids Research, 17, 9494 (1989)], λZAP II (производство Stratagene Corporation), λgt 10, λgt 11 [DNA Cloning: A Practical Approach, I, 49 (1985)], Lambda BlueMid (производство Clontech Laboratories, Inc.), λExCell, pT7T3-18U (производство Pharmacia Corporation), pcD2 [Mol. Cell. Biol., 3, 280 (1983)], pUC18 [Gene, 33, 103 (1985)] или т.п.
В качестве E. Coli, в которые вводят созданную с фаговым или плазмидным вектором библиотеку кДНК, можно использовать любые E. coli, способные включить, экспрессировать и сохранять кДНК библиотеки. Например, можно использовать такие штаммы, как XL1-Blue MRF’ [Strategies, 5, 81 (1992)], C600 [Genetics, 39, 440 (1954)], Y1088, Y1090 [Science, 222, 778 (1983)], NM522 [J. Mol. Biol., 166, 1 (1983)], K802 [J. Mol. Biol., 16, 118 (1966)], JM105 [Gene, 38, 275 (1985)] или подобные указанным.
Для того, чтобы из библиотеки кДНК отобрать клоны кДНК, кодирующей VH или VL антитела не человеческого происхождения, применяют метод гибридизации колоний с использованием радиоактивно либо флуоресцентно меченных зондов или гибридизацию бляшек [Molecular Cloning, A Laboratory Manual, 2-е издание, Cold Spring Harbor Laboratory Press (1989)] или подобные методы.
Кроме того, можно получить кДНК, кодирующую VH или VL, можно также получать путем полимеразной цепной реакции (далее обозначается ПЦР), подобрав соответствующие праймеры [Molecular Cloning, A Laboratory Manual, 2-е издание, Cold Spring Harbor Laboratory Press (1989), Current Protocols in Molecular Biology, дополнение 1, John Wiley & Sons (1987-1997)] и используя кДНК, синтезированную по мРНК, или библиотеку кДНК, в качестве матрицы.
Отобранные кДНК расщепляют подходящими ферментами рестрикции или побобным, затем клонируют в плазмиде, например в pBluescript SK(-) (производство Stratagene Corporation), и определяют нуклеотидную последовательность кДНК обычно применяемыми методами анализа нуклеотидных последовательностей. Например, используют терминирующие дидезоксинуклеотиды [Proc. Natl. Acad. Sci. USA, 74, 5463 (1977)] или проводят автоматическое определение нуклеотидной последовательности, например с помощью секвенатора A.L.F. DNA sequencer (производство Pharmacia Corporation).
По установленной полной нуклеотидной последовательности выводят полную аминокислотную последовательность VH или VL и сравнивают ее с полной аминокислотной последовательностью соответствующей VH и VL известного антитела [Sequences of Proteins of Immunological Interest, US Dept. Health and Human Services (1991)]; таким образом выясняют, кодирует ли полученная кДНК полноразмерную аминокислотную последовательность VH или VL антитела, содержащую сигнальный пептид, необходимый для секреции.
Располагая полными аминокислотными последовательностями VH и VL антитела, содержащими последовательность сигнала секреции, сравнивают полную аминокислотную последовательность с VH и VL известного антитела [Sequences of Proteins of Immunological Interest, US Dept. Health and Human Services (1991)] и таким образом устанавливают длину последовательности сигнала секреции, , и N-концевой аминокислотной последовательности, а также к какой подгруппе они принадлежат.
Кроме того, путем сравнения последовательностей VH и VL с последовательностями известного анитела можно установить аминокислотные последовательности CDR в VH и VL [Sequences of Proteins of Immunological Interest, US Dept. Health and Human Services (1991)].
Также, располагая полученными полными аминокислотными последовательностями VH и VL, можно выяснить, являюттся ли указанные последовательности VH и VL новыми, например проведя поиск гомологии, например, с помощью компьютерных программ BLAST [J. Mol. Biol., 215, 403 (1990)] или подобных, в таких базах данных, как SWISS-PROT или PIR-Protein..
(2) Конструирование экспрессионного вектора для получения
генно-инженерного рекомбинантого антитела
Экспрессионный вектор для получения генно-инженерного можно получить путем клонирования ДНК, кодирующую по меньшей мере СН или СL человеческого антитела, в экпресиоонном векторе, подходящим для использования в животной клетке.
В качестве С областей можно исвпользовать СН и СL произвольного человеческого антитела, например СН подкласса γ1 и CL подкласса κ человеческого антитела или им подобные. В качестве ДНК, кодирующей СН или СL человеческого антитела используют кДНК, но пригодна также хромосомная ДНК, в которой имеются экзоны и интроны.
В качестве экспрессионного вектора для животных клеток можно использовать любой вектор, при условии, что он может включить ген, кодирующий С область человеческого антитела и экпрессировать ее; например, пригодны векторы pAGE107 [Cytotechnol., 3, 133 (1990)], pAGE103 [J. Biochem., 101, 1307 (1987)], pHSG274 [Gene, 27, 223 (1984)], pKCR [Proc. Natl. Acad. Sci. USA, 78, 1527 (1981)], pSG1bd2-4 [Cytotechnol., 4, 173 (1990)], pSE1UK1Sed1-3 [Cytotechnol., 13, 79 (1993)], INPEP4 (производство Biogen-IDEC, Inc.), N5KG1val (патент США № 6,001,358), N5KG4PE R409K (описан в публикации WO 2006/033386), векторы N5KG2 (описаны в публикации WO 2003/033538), транспозонные векторы (WO 2010/143698) и т.п.
Промотором и энхансером в экспрессионном векторе для животных клеток могут служить ранний промотор SV40 [J. Biochem., 101, 1307 (1987)], (LTR) вируса лейкоза мышей Молони [Biochem. Biophys. Res. Commun., 149, 960 (1987)], промотор CMV (патент CША № 5,168,06) или промотор [Cell, 41, 479 (1985]) и энхансер [Cell, 33, 717 (1983)] гена H-цепи иммуноглобулина или подобные им регуляторные элементы.
Для экспрессии генно-инженерного рекомбинантного антитела в целях облегчения конструирования вектора, его введения в животные клетки, равновесного соотношения между уровнями экспрессии H- и L- цепей антитела и из других соображений используют вектор, содержащий гены как L-, так и H-цепи (вектор тандемного типа) [J. Immunol. Methods, 167, 271 (1994)], но можно использовать и комбинацию из нескольких векторов, каждый из которых несет один ген – H- либо L-цепи ( векторы)
Для получения генно-инженерных рекомбинантных антител используют такие экспрессионные векторы тандемного типа, как pKANTEX93 (WO 97/10354), pEE18 [Hybridoma, 17, 559 (1998)], N5KG1val (патент CША №6,001,358), N5KG4PE R409K (описан в публикации WO 2006/033386), векторы N5KG2 (описаны в публикации WO 2003/033538), транспозонный вектор Tol2 (WO 2010/143698) или подобные им.
(3) Конструирование экспрессионного вектора для получения
химерного антитела
Чтобы сделать экспрессионный вектор для получения химерного антитела, кДНК, кодирующую VH или VL антитела не человеческого происхождения, полученную в (1), клонируют в экспрессионном векторе для получения рекомбинантного антитела, сконструированного в (2), расположив ее перед каждым из генов, кодирующих СН или СL человеческого антитела.
Сначала, чтобы лигировать кДНК, кодирующую VH или VL антитела не человеческого происхождения, со стороны 3’-конца, с последовательностями СН или СL человеческого антитела, располагающимися на 5’-конце, получают кДНК, кодирующие VH и VL и сконструированные так, что нуклеотидная последовательность участка лигирования кодирует соответственные аминокислоты и имеется последовательность, узнаваемую подходящим ферментом рестрикции. Затем каждую из полученных кДНК VH и VL клонируют в экспрессионном векторе для получения генно-инженерного рекомбинантного антитела, сконструрованном в (2), выше гена, кодирующего СН или СL человеческого антитела соответственно, так что они экспрессируются в нужной форме; таким образом получается экспрессионный вектор для химерного антитела.
Кроме того, каждую из кДНК, кодирующих VH или VL антитела не человеческого происхождения, амплифицируют путем ПЦР, используя синтетическую ДНК, содержащую на обоих концах соответствующую нуклеотидную последовательность, распознаваемую ферментом рестрикции, и клонируют в экспрессионном векторе для получения генно-инженерного рекомбинантного антитела, сконструированном в (2); таким образом тоже получается экспрессионный вектор для химерного антитела.
(4) Получение кДНК, кодирующей V область
гуманизированного антитела
кДНК, кодирующую VH или VL гуманизированного антитела, получают следующим образом. Сначала выбирают те аминокислотные последовательности каркасных участков (далее обозначаемых (FR)) VH или VL человеческого антитела, к которым должны быть «привиты» аминокислотные последовательности CDR VH или VL не человеческого антитела, полученного в (1).
В качестве аминокислотной последовательности FR можно выбрать любую аминокислотную последовательность, происходящую из человеческого антитела. Например, можно использовать аминокислотную последовательность FR человеческого антитела, зарегистрированную в такой базе данных, как Protein Data Bank, или аминокислотную последовательность, типичную для данной подгруппы FR человеческих антител [Sequences of Proteins of Immunological Interest, Министерство здравоохранения и социального обеспечения США (1991)] и т.п. Чтобы избежать снижения активности связывания антитела с антигеном, выбирают аминокислотные последовательности FR человеческих антител, как можно более гомологичные (со степенью гомологии 60% или более) аминокислотным последовательностям соответствующих FR в VH или VL исходного не человеческого антитела.
Затем каждую из аминокислотных последовательностей CDR исходного не человеческого антитела «прививают» в выбранную аминокислотную последовательности FR в VH или VL человеческого антитела и конструируют аминокислотные последовательности VH или VL гуманизированного антитела. По составленной аминокислотной последовательности выводят нуклеотидную последовательность с учетом предпочтения кодонов, свойственную нуклеотидной последовательности гена данного антитела [Sequences of Proteins of Immunological Interest, Министерство здравоохранения и социального обеспечения США (1991)], и составляют последовательность кДНК для VH или VL гуманизированного антитела.
На основании составленной последовательности кДНК синтезируют несколько ДНК длиной 100-150 нуклеотидов и, используя их, проводят ПЦР. В таком случае ради эффективности реакции ПЦР и с учетом длины синтезируемой ДНК для каждой тяжелой и для каждой легкой цепи получают 4-6 синтетических ДНК. Также возможно синтезировать и использовать синтетическую ДНК, кодирующую полноразмерную вариабельную область.
Далее встраивают нуклеотидную последовательность, распознаваемую подходящим ферментом рестрикции, на 5’-конце синтетической ДНК с двух сторон, что позволяет клонировать кДНК, кодирующую VH или VL гуманизированного антитела в экспрессионном векторе для получения генно-инженерного рекомбинантного антитела по у (2). После проведения ПЦР каждый амплифицированный продукт клонируют в плазмидном векторе, например pBluescript SK(-) (производство Stratagene Corporation), определяют нуклеотидную последовательность, как описано в (1), и таким образом получают плазмиду, содержащую ДНК, кодирующую аминокислотную последовательность VH или VL желаемого гуманизированного антитела.
(5) Модификация аминокислотной последовательности
V области гуманизированного антитела
Активность связывания антигена у гуманизированного антитела, полученного путем «прививки» только CDR VH и VL антитела не человеческого присхождения к каркасным участкам VH и VL человеческого антитела, ниже, чем у исходного не человеческого антитела. [BIO/TECHNOLOGY, 9, 266 (1991)]. В связи с этим сниженную активность связывания антигена такого гуманизированного антитела можно повысить ; для этого нужно установить, какие аминокислотные остатки в последовательностях FR участков VH или VL человеческого антитела непосредственно участвуют в связывании антигена, какие взаимодействуют с CDR и какие обеспечивают должную конформацию молекулы антитела и участвуют в связывании антигена косвенным образом, а на основании этого заменить эти аминокислотные остатки на те, которые им соответствуют в полипептидных цепях антитела не человеческого происхождения.
Чтобы идентифицировать аминокислотные остатки FR, участвующие в связывании антигена, воссоздают и анализируют пространственную структуру молекулы антитела путем рентгеновской кристаллографии [J. Mol. Biol., 112, 535 (1977)] или компьютерного моделирования [Protein Engineering, 7, 1501 (1994)] или иными методами. Также можно получить модифицированное гуманизированное антитело с нужной активностью связывания антигена путем проб и ошибок: получить по нескольку различных модификаций для каждого антитела и проверить, как связаны данные модификации с активностью связывания антигена.
Аминокислотные остатки FR участков VH или VL человеческого антитела могут быть заменены путем проведения ПЦР, описанной в (4); при этом для изменений используется синтетическая ДНК. После ПЦР определяют нуклеотидную последовательность продукта амплификации, как описано в (1), чтобы убедиться, что желаемые изменения произошли.
(6) Конструирование экспрессионного вектора
для получения гуманизированного антитела
Экспрессионный вектор для получения гуманизированного антитела конструируют путем клонирования каждой из кДНК, кодирующих VH или VL составленного гуманизированного антитела, перед соответствующими генами, кодирующими CH или CL человеческого антитела, в экспрессионном векторе для получения генно-инженерного рекомбинантного антитела по пункту (2).
Например, в экспрессионном векторе для получения генно-инженерного рекомбинантного антитела по пункту (2) перед каждым из генов, кодирующих CH или CL человеческого антитела, встраивают нуклеотидную последовательность, распознаваемую подходящим ферментом рестрикции, на 5`-конце синтетической ДНК с двух сторон из числа синтетических ДНК, использовавшихся при конструировании VH или VL гуманизированного антитела, полученного по пункту (4) или (5); в результате они экспрессируются в должной форме.
(7) Конструирование экспрессионного вектора
для получения человеческого антитела
Когда получают гибридому, продуцирующая моноклональное антитело, используя в качестве иммунизированного животного, тех, у того образуются человеческие антитела, возможно установить аминокислотные последовательности и последовательности кДНК для VH и VL человеческого антитела по (1). И тогда можно сконструировать экспрессионный вектор для получения человеческого антитела, клонируя каждый из генов, кодирующих VH или VL человеческого антитела, полученного в (1), перед каждым из генов, кодирующих CH или СL человеческого антитела в экспрессионном векторе для получения генно-инженерного рекомбинантного антитела, полученном по (2).
(8) Временная экспрессия генно-инженерного рекомбинантного антитела
Активность связывания антигена многими различными генно-инженерными рекомбинантными антителами можно с успехом определять, осуществляя временную экспрессию генно-инженерного рекомбинантного антитела с использованием экспрессионного вектора для получения генно-инженерного рекомбинантного антитела, полученного в (3), (6) и (7) или экспрессионного вектора, полученного в результате модификации указанного вектора.
В качестве клеток-хозяев для введения экспрессионного вектора пригодны любые клетки, способные к экспрессии генно-инженерного рекомбинантного антитела, например клетки COS-7 (регистрационный номер CRL1651 в Американской коллекции типовых культур (ATCC)). Для введения экспрессионного вектора в клетки COS-7 применяется трансфекция с использованием DEAE-декстрана [Methods in Nucleic Acids Res., CRC press (1991)], липофекция [Proc. Natl. Acad. Sci. USA, 84, 7413 (1987)] и другие подобные методы.
После введения экспрессионного вектора в клетки-хозяева в супернатанте культуры определяют уровень экспрессии генно-инженерного рекомбинантного антитела и его активность связывания антигена, применяя иммуноферментный анализ [Monoclonal Antibodies-Principles and practice, 3-е издание, Academic Press (1996), Antibodies-A Laboratory Manual, Cold Spring Harbor Laboratory (1988), Monoclonal Antibody Experimental Manual, Kodansha scientific books (1987)] или иной метод.
(9) Получение линии клеток со стабильной экспрессией
генно-инженерного рекомбинантного антитела и получение генно-инженерного рекомбинантного антитела
Трансформированные клетки со стабильной экспрессией генно-инженерного рекомбинантного антитела получают путем введения экспрессионного вектора для получения генно-инженерного рекомбинантного антитела по (3), (6) и (7) в подходящие клетки-хозяева.
Для введения экспрессионного вектора в клетки-хозяева применяют, например, электропорацию [JP- A-H2-257891, Cytotechnology, 3, 133 (1990)], обработку ионами кальция, а также используют сферопласты, ацетат лития, фосфат кальция, липофекцию и другие подобные методы. Кроме того, можно вводить нужный ген в организм животного (см. ниже), например, путем микроинъекций; способа введения гена в ES-клетки путем электропорации или липофекции, а также переноса ядер и другими методами.
В качестве клеток-хозяев, в которые вводят экспрессионный вектор для получения генно-инженерного рекомбинантного антитела, могут служить любые клетки, способные к экспрессии генно-инженерного рекомбинантного антитела. Например, пригодны мышиные клетки ES SP2/0-Ag14 (ATCC CRL 1581), P3X63-Ag8.653 (ATCC CRL 1580), клетки китайского хомячка CHO-K1 (ATCC CCL-61), DUKXB11 (ATCC CCL-9096), Pro-5 (ATCC CCL-1781), CHO-S (Life Technologies, Cat No. 11619), CHO с дефицитом дигидрофолат-редуктазы (CHO/DG44) [Proc. Natl. Acad. Sci. USA, 77, 4216 (1980)], СНО-Lec13 с приобретенной устойчивостью к лектину [Somatic Cell and Molecular Genetics, 12, 55 (1986)], CНО с дефицитом α1,6-фукозилтрансферазы (WO 2005/035586, WO 02/31140), крысиные клетки YB2/3HL.P2.G11.16Ag.20 (ATCC CRL 1662) и другие подобные клетки.
Также можно использовать клетки-хозяева, в которых понижена или утрачена активность какого-либо специфического белка, например фермента, участвующего во внутриклеточном синтезе сахарного произхводного нуклеотида GDP-фукозы; или фермента, участвующего в модификации сахарной цепи, например обеспечивающего связь α1-6 между молекулами фукозы и N-ацетилглюкозамина на восстанавливающем конце N-гликозидной углеводной цепи; или белка, участвующего в транспорте GDP-фукозы в аппарате Гольджи, или другие подобные белки; например, пригодны клетки СНО с дефицитом α1,6-фукозилтрансферазы (WO 2005/035586, WO 02/31140).
После введения экспрессионного вектора в клетки-хозяева отбирают трансформанты со стабильной экспрессией генно-инженерного рекомбинантного антитела путем культивирования полученных трансформированных клеток в среде для культур животных клеток, содержащей лекарственное средство, например G418 сульфат (JP- A-H2-257891).
Для культивирования животных клеток по данному изобретению используются такие среды, как RPMI 1640 (производство Invitrogen, Inc.), GIT (производство Nippon Pharmaceutical Co., Ltd.), EX-CELL 301 (производство JRH Biosciences, Inc.), EX-CELL 302 (производство JRH Bioscience, Inc.), EX-CELL 325 (производство JRH Bioscience., Inc.), IMDM (производство Invitrogen, Inc.) или Hybridoma-SFM (производство Invitrogen, Inc.), или эти среды с добавлением различных ингредиентов, например FBS, или другие подобные среды. Отобранные трансформанты культивируют в подходящей среде, в клетках экспрессируется генно-инженерное рекомбинантное антитело, которое накапливается в культуральной среде. В супернатанте культуральной среды количественно определяют уровень экспрессии генно-инженерного рекомбинантного антитела и его активность связывания антигена, применяя ELISA или другие методы. Кроме того, экспрессию генно-инженерного рекомбинантного антитела в трансформированных клетках можно усилить, если использовать систему амплификации DHFR,) (JP- A-H2-257891) или подобную.
Для очистки генно-инженерного рекомбинантного антитела из супернатанта культуры трансформированных клеток используют колонки с протеином А [Monoclonal Antibodies - Principles and Practice, Third Edition, Academic Press (1996), Antibodies - A Laboratory Manual, Cold Spring Harbor Laboratory (1988)]. Также для очистки можно сочетать такие методы очистки белков, как гель-фильтрация, ионообменная хроматография и ультрафильтрация.
Для определения молекулярной массы H- и L-цепей и цельной молекулы очищенного генно-инженерного рекомбинантного антитела применяют электрофорез в полиакриламидном геле [Nature, 227, 680 (1970)], или вестерн-блоттинг [Monoclonal Antibodies - Principles and Practice, Third Edition, Academic Press (1996), Antibodies - A Laboratory Manual, Cold Spring Harbor Laboratory (1988)], или другие методы.
3. Дизайн биспецифических антител
Биспецифическое антител по данному изобретению может быть создано путем конструирования каждого из полипептидов N-концевой стороны и часть IgG, и конструирования биспецифического антитела, где они объединены.
3-1. Конструирование части IgG
Для того, чтобы сделать часть IgG, получают моноклональные антитела способом, описанным выше в 1; для каждого антитела определяют последовательности кДНК, кодирующих CDR и вариабельную область так, как описано выше в 2, и конструируют часть IgG, включающую CDR или вариабельную область антитела.
3-2. Конструирование полипептида N-концевой стороны
Когда CDR или вариабельная область антитела входят в состав полипептида N-концевой стороны, для его получения определяют нуклеотидные последовательности, кодирующие CDR или вариабельную область антитела согласно способу, как описано выше в 1 и 2, и конструируют полипептид N-концевой стороны, включающий их. Полипептид N-концевой стороны может быть одноцепочечным, в котором VH и VL соединены непосредственно или посредством соответствующего линкера в scFV или подобную структуру; или экспрессироваться в двухцепочечной форме, например, Fab, dsFv и подобные им структуры, где связь S-S образуется после экспрессии; или иметь структуру типа VHH, или подобную ей. У полученных вариантов полипептида N-концевой стороны описанными выше методами определяют активность связывания антигена и выбирают тот, который обладает ею в должной степени.
4. Получение биспецифических антител
4-1. Получение биспецифического антитела с легкими цепями общего типа
Биспецифическое антитело, в котором полипептидом N-концевой стороны является Fab и легкая цепь имеет структуру, обычную для Fab и части IgG, получают следующим образом.
Синтезируют ДНК, кодирующую полипептид, в котором VH-CH1 полипептида N-концевой стороны соединен с VH части IgG, а также ДНК, кодирующие VL каждого из антител; эти ДНК встраивают в экспрессионный вектор для получения генно-инженерного рекомбинантного антитела, описанный выше в 2. (2), и осуществляют экспрессию, получая таким образом биспецифическое антитело. В том случае, когда часть IgG и полипептид N-концевой стороны соединены посредством линкера, синтезируют также ДНК, кодирующую линкер.
4-2. Получение биспецифического антитела в котором полипептид N-концевой стороны включает легкие цепи необщего типа, содержащие CDR
(1) Биспецифическое антитело с легкими цепями необщего типа, в котором полипептидом N-концевой стороны является Fab, получают следующим образом.
Синтезируют ДНК, кодирующую полипептид, в котором VL-CL полипептида N-концевой стороны соединена VH части IgG, а также ДНК, кодирующую VH-СН1 полипептида N- концевой стороны и ДНК, кодирующую VL части IgG; эти ДНК включают в экспрессионный вектор для получения генно-инженерного рекомбинантного антитела, описанный выше в 2. (2), и осуществляют экспрессию, получая таким образом биспецифическое антитело. В том случае, когда часть IgG и полипептид N-концевой стороны соединены посредством линкера, синтезируют также ДНК, кодирующую линкер.
(2) Биспецифическое антитело, в котором полипептидом N-концевой стороны является VHH, получают следующим образом.
Синтезируют ДНК, кодирующую полипептид, в котором соединены VHH и VH части IgG, а также ДНК, кодирующую VL части IgG; эти ДНК включают в экспрессионный вектор для получения генно-инженерного рекомбинантного антитела, описанный выше в 2. (2) и осуществляют экспрессию, получая таким образом биспецифическое антитело. В том случае, когда VHH и часть IgG соединены посредством линкера, синтезируют также ДНК, кодирующую линкер.
(3) Биспецифическое антитело, в котором полипептидом N-концевой стороны является полипептид, включающий scFv, dsFv или CDR и отличный от описанных выше в (1) и (2), получают следующим образом.
Если полипептид N-концевой стороны является одноцепочечным, то синтезируют ДНК, в которой ДНК, кодирующая полипептид N-концевой стороны, соединена с ДНК, кодирующей VH части IgG. Если полипептид N-концевой стороны представляет собой комплекс, состоящий из двух одноцепочечных полипептидов, то синтезируют ДНК, кодирующую один из одноцепочечных полипептидов, образующих полипептид N-концевой стороны, которая соединена с ДНК, кодирующей VH части Ig, а также ДНК, кодирующую другой одноцепочечный полипептид, образующий полипептид N-концевой стороны. Кроме того, синтезируют ДНК, кодирующую VL части IgG. В том случае, когда часть IgG и полипептид N-концевой стороны соединены посредством линкера, синтезируют также ДНК, кодирующую линкер. Эти ДНК включают в экспрессионный вектор для получения генно-инженерного рекомбинантного антитела, описанный выше в 2. (2), и осуществляют экспрессию, получая таким образом биспецифическое антитело.
4-3. Получение биспецифического антитела, включающего полипептид N-концевой стороны, отличный от описанных выше
Когда N-концевой полипептид отличен от описанных выше, биспецифическое антитело по данному изобретению получают следующим образом.
Если N-концевой полипептид является одноцепочечным, то синтезируют ДНК, в которой соединены ДНК, кодирующая полипептид N-концевой стороны, и ДНК, кодирующая VH части IgG. Если N-концевой полипептид представлен комплексом, состоящим из двух или более одноцепочечных полипептидов, то синтезируемая ДНК, кодирующая один из одноцепочечных полипептидов, образующих полипептид N-концевой стороны, соединена с ДНК, кодирующей VH части IgG, а также синтезируют ДНК, кодирующую остальные одноцепочечные полипептиды, образующие полипептид N-концевой стороны. Кроме того, синтезируют ДНК, кодирующую VL части IgG. В том случае, когда часть IgG и полипептид N-концевой стороны соединены посредством линкера, синтезируют также ДНК, кодирующую линкер. Эти ДНК включают в экспрессионный вектор для получения генно-инженерного рекомбинантного антитела, описанный выше в разделе 2.(2), и осуществляют экспрессию, получая таким образом биспецифическое антитело.
Эти ДНК включают в экспрессионный вектор для получения генно-инженерного рекомбинантного антитела, описанный выше в 2. (2), и осуществляют экспрессию, получая таким образом биспецифическое антитело. В том случае, когда часть IgG и полипептид N-концевой стороны соединены посредством линкера, синтезируют также ДНК, кодирующую линкер.
Домен, связывающий антиген, можно получить и выделить такими методами, как фаговый или дрожжевой дисплей, отличными от метода с использованием гибридом, описанного выше в 1 [Emmanuelle Laffy et. al., Human Antibodies 14, 33-55, (2005)].
В тех случаях, когда нужно получить биспецифическое антитело, состоящее из нескольких VH и одной VL (далее в настоящем документе такое антитело называется биспецифическим антителом с легкой цепью общего типа), проводят скрининг методом фагового дисплея или подобным способом и отбирают VH цепи, наиболее подходящие для данной VL, так что в получаемом биспецифическом антителе каждый антиген-связывающий домен будет взаимодействовать с определенным антигеном..
А именно, сначала иммунизируют животное первым антигеном, как описано выше в 1, и получают гибридомы из селезенки иммунизированного животного; затем клонируют нуклеотидную последовательность, кодирующую первый антиген-связывающий домен. После этого животное иммунизируют вторым антигеном и получают из его селезенки библиотеку кДНК; затем с помощью ПЦР из этой библиотеки получают ДНК, кодирующую аминокислотную последовательность VH.
После этого получают фаговую библиотеку, экспрессирующую scFv, в которых соединены VH, полученная в результате иммунизации вторым антигеном, и VL первого антиген-связывающего домена,; путем пэннинга фаговой библиотеки отбирают фаги, экспонирующие scFv, специфично связывающимися со вторым антигеном. Используя отобранный фаг, клонируют ДНК, кодирующую аминокислотную последовательность VH второго антиген-связывающего домена.
Если полипептид N-концевой стороны представлен Fab, то конструируют ДНК, кодирующую аминокислотную последовательность полипептида, в котором VH первого антиген-связывающего домена, и VH второго антиген-связывающего домена, соединены посредством участка CH1 или CH1 и линкера; эту ДНК и ДНК, кодирующую аминокислотную последовательность одиночной VL встраивают, например, в экспрессионный вектор для получения генно-инженерного рекомбинантного антитела, описанного выше в 2. (2) таким образом получается экспрессионный вектор для биспецифического антитела по данному изобретению.
5. Определение активности биспецифических антител или фрагментов данного антитела по хизобретению
Определение активности очищенных биспецифических антител или биспецифически фрагментов данного антитела проводят следующим образом.
Активность связывания биспецифических антител по данному изобретению с клетками линии, характеризующейся экспрессией по меньшей мере TfR, или антигена клеточной поверхности, например EGFR, или обоих этих веществ, можно измерить с помощью аналитической системы связывания, описанной выше в 1. (7).
Активность CDC или активность ADCC, направленную против клеток, в которых экспрессируется по меньшей мере TfR , или антиген клеточной поверхности, например EGFR, или оба этих вещества, определяют известными в данной области техники методами измерения [Cancer Immunol. Immunother. 36, 373 (1993)].
«Противоклеточную» активность (называемую также активностью подавления роста)биспецифических антител по данному изобретению, можно измерить следующим способом. Например, высевают клетки на 96-луночный планшет, добавляют антитела и культивируют клетки некоторое время, после чего прибавляют реагент WST-8 (производство Dojindo Molecular Technologies, Inc.), дают совершиться реакции и измеряют поглощение на длине волны 450 нм с помощью планшетного ридера; полученные данные позволяют оценить жизнеспособность клеток.
Передачу сигнала внутрь клетки с TfR или антигена клеточной поверхности, например, EGFR, определяют путем выявления фосфорилирования белков в клетке методом вестерн-блоттинга или подобным способом.
6. Способ лечения заболеваний с использованием биспецифических антител или фрагментов данного антитела по изобретению
Биспецифические антитела или биспецифические фрагменты антител по данному изобретению можно использовать для лечения заболеваний, связанных по меньшей мере с TfR, или с антигеном клеточной поверхности, например EGFR, или с обоими этими веществами; предпочтительно это касается заболеваний, в которых играют роль клетки, где экспрессируются TfR и антиген клеточной поверхности, например EGFR. Примером заболеваний, связанных по меньшей мере с TfR, или с антигеном клеточной поверхности, например EGFR, или с обоими этими веществами, являются злокачественные опухоли, раковые заболевания и т.п.
Примеры злокачественных опухолей и раковых заболеванийвключают рак толстой кишки, колоректальный рак, рак легких, рак молочной железы, глиому, злокачественную меланому, рак щитовидной железы, почечноклеточную карциному, лейкозы, лимфомы, Т-клеточную лимфому, рак желудка, рак поджелудочной железы, рак шейки матки, рак тела матки, рак яичника, рак желчного протока, рак пищевода, рак печени, рак головы и шеи, плоскоклеточный рак, рак кожи, раковые поражения мочевых путей, рак мочевого пузыря, рак предстательной железы, хориокарциному, фарингеальный рак, рак гортани, опухоли плевры, арренобластому, гиперплазию эндометрия, эндометриоз, эмбриому, фибросаркому, саркому Капоши, ангиому, пещеристую гемангиому, ангиобластому, ретинобластому, астроцитому, нейрофиброму, олигодендроглиому, медуллобластому, нейробластому, глиому, рабдомиосаркому, глиобластому, остеогенную саркому, лейомиосаркому, опухоль Вильмса и проч.
Терапевтический агент, содержащий биспецифическое антитело или биспецифический фрагмент антитела по данному изобретению, или их производные, может содержать только указанное антитело, или фрагмент антитела , или их производные в качестве активного ингредиента, однако, как правило, предпочтителен терапевтический агент, представленный фармацевтическим препаратом, полученным известными в области фармацевтики методами, предполагающими смешивание активного ингредиента с одним или более фармакологически приемлемыми носителями.
Примеры путей введения терапевтического агента по данному изобретению включают пероральное или парентеральное введение, например введение в ротовую полость, в воздухоносные пути, в прямую кишку, подкожно, внутримышечно и внутривенно. Примеры лекарственных форм терапевтического агента по данному изобретению включают аэрозоли и спреи, капсулы, таблетки, порошки, гранулы, сиропы, эмульсии, суппозитории, инъекционные препараты, мази, пластыри и проч. Различные фармацевтические препараты терапевтического агента по данному изобретению могут быть получены обычными для таких целей методами, включающими использование эксципиентов, наполнителей, связующих агентов, смачивающих/увлажняющих агентов, дезинтегрирующих агентов, поверхностно-активных веществ, агентов, улучшающих скольжение, диспергирующих агентов, забуферивающих агентов, консервантов, солюбилизирующих агентов, антисептиков, красителей, ароматизаторов, стабилизирующих агентов и проч.
Примеры эксципиентов включают лактозу, фруктозу, глюкозу, кукурузный крахмал, сорбит, кристаллическую целлюлозу, стерильную воду, этиловый спирт, глицерин, физиологический солевой раствор, буферные растворы и проч. Примеры дезинтегрирующих агентов включают крахмал, альгинат натрия, желатин, карбонат кальция, цитрат кальция, декстрин, карбонат магния, синтетический силикат магния и проч.
Примеры связующих агентов включают метилцеллюлозу или ее соли, этилцеллюлозу, аравийскую камедь, желатин, гидроксипропилцеллюлозу, поливинилпирролидон и др. Примеры агентов, улучшающих- скольжение, включают тальк, стеарат магния, полиэтиленгликоль, гидрогенизированное растительное масло и др.
Примеры стабилизирующих агентов включают аминокислоты, например аргинин, гистидин, лизин и метионин, человеческий сывороточный альбумин, желатин, декстран 40, метилцеллюлозу, сульфит натрия, метасульфит натрия и др.
Примеры других дополнительных ингредиентов включают сироп, вазелин, глицерин, этиловый спирт, пропиленгликоль, лимонную кислоту, хлорид натрия, нитрит натрия, фосфат натрия и др.
Примеры фармацевтических препаратов, пригодных для перорального применения, включают эмульсии, сиропы, капсулы, таблетки, порошки, гранулы и др.
При изготовлении жидких фармацевтических препаратов, например эмульсий или сиропов, используется вода; сахара , например сахароза, сорбит или фруктоза; гликоли, например полиэтиленгликоль или пропиленгликоль; масла, например кунжутное, оливковое или соевое масло; консерванты, например эфир пара-гидроксибензойной кислоты; ароматизаторы, например клубничный или мятный; и другие добавки.
Для изготовления капсул, таблеток, порошков, гранул и подобных препаратов используются эксципиенты, например лактоза, глюкоза, сахароза или маннит; дезинтегрирующие агенты, например крахмал или альгинат натрия; агенты, улучшающие скольжение, например стеарат магния или тальк; связующие агенты, например поливиниловый спирт, гидроксипропилцеллюлоза или желатин; поверхностно-активные вещества, например эфиры жирных кислот; пластифицирующие агенты, например глицерин; и другие подобные добавки.
Примеры фармацевтических препаратов, пригодных для парентерального введения, включают инъекционные препараты, суппозитории, спреи и др. Для изготовления инъекционных препаратов используются носители, представленные солевым раствором, раствором глюкозы или смесью этих двух растворов.
Для изготовления суппозиториев используют такие носители, как масло какао, гидрогенизированные жиры или карбоновые кислоты. Для изготовления спреев используют носители, не оказывающие раздражающего действия на слизистую оболочку ротовой полости или воздухоносных путей пациента и диспергирующие моноклональные антитела или фрагменты антитела по данному изобретению в виде мелких частиц. что способствует их поглощению, и др. Примеры таких носителей включают лактозу, глицерин и др. Также терапевтический агент по данному изобретению может быть представлен препаратом в форме аэрозоля или сухого порошка. Кроме того, некоторые компоненты из числа добавок, используемых в фармацевтических препаратах для перорального применения, могут также использоваться в указанных выше препаратах, вводимых парентерально.
Эффективная дозировка препарата по данному изобретению, складывающаяся из эффективного количества биспецифических антител и нужного количества разбавителя и фармакологически приемлемого носителя, составляет от 0,0001 мг на 1 кг массы тела до 100 мг/кг на один прием, причем промежуток времени между введениями составляет от 2 суток до 8 недель.
7. Способ диагностики заболеваний с использованием биспецифических антител или биспецифически фрагментов антител по данному изобретению
Выявление или количественное определение клеток, в которых экспрессируется по меньшей мере TfR, или антиген клеточной поверхности, например EGFR, или оба этих вещества, с использованием биспецифических антител или биспецифических фрагментов антител по данному изобретению позволяет диагностировать заболевания, связанные по меньшей мере с TfR, или с антигеном клеточной поверхности, например EGFR, или с обоими этими веществами, предпочтительно заболеваний, в которых играют роль клетки, где эксспрессируются TfR и антиген клеточной поверхности, например EGFR.
Диагностирование таких заболеваний, как злокачественные опухоли и раковые заболевани, которые связаны по меньшей мере сTfR, или с антигеном клеточной поверхности, например EGFR, или с обоими этими веществами, может осуществляться, например, путем выявления или количественного определения TfR и/или антигена клеточной поверхности, например EGFR, следующим образом.
Вначале исследуют биологические образцы от множества здоровых индивидов; для выявления или количественного определения в них TfR и/или антигена клеточной поверхности, например EGFR, с помощью описанных ниже иммунологических методов анализа с использованием биспецифических антител, или биспецифических фрагментов антител по данному изобретению, или их производных; анализируют полученные данные о количестве TfR и/или антигена клеточной поверхности, например EGFR, в биологических образцах здоровых индивидов.
Затем теми же методами определяют количество TfR и/или антигена клеточной поверхности, например EGFR, в биологическом образце тестируемого субъекта и сравнивают полученные данные с таковыми здоровых индивидов. Если у тестируемого суюъекта количество TfR и/или антигена клеточной поверхности, например, EGFR, повышено по сравнению со здоровыми индивидами, то у него диагностируют рак. Таким же образом диагностируют другие заболевания, связанные с TfR и/или с антигеном клеточной поверхности, например EGFR.
Иммунологические методы – это методы выявления или количественного определения антитела или антигена при помощи меченых антигена или антитела соответственно. Примеры таких методов включают анализ, в котором используют антитела, меченные радиоактивными изотопами; иммуноферментный анализ; анализ с использованием флуоресцентных или люминесцентных меток, вестерн-блоттинг, физико-химические методы и др.
Примеры анализа с использованием радиоактивно меченных антител включают метод, в котором биспецифическое антитело или биспецифический фрагмент антитела по данному изобретению взаимодействует с антигеном, или с клеткой, где экспрессируется антиген, или с подобным, затем взаимодействует с анти-иммуноглобулиновым антителом или его связывающим фрагментом, несущим радиоактивную метку, и выявляется с помощью сцинтилляционного счетчика или подобного измерительного прибора.
Примеры иммуноферментного анализа включают метод, в котором биспецифическое антитело или биспецифический фрагмент антитела по данному изобретению взаимодействует с антигеном, или с клеткой, где экспрессируется антиген, или с подобным, затем взаимодействует с анти-иммуноглобулиновым антителом или его связывающим фрагментом, несущим метку, и с последующим измерением окраса с помощью абсорбциометра. Примером такого метода является сэндвич-вариант ELISA.
В качестве меток для иммуноферментного анализа используют известные ферментные метки [IGAKU-SHOIN Ltd. (1987)], например, щелочную фосфатазу, пероксидазу, люциферазу; используется также биотиновая метка и др.
В сэндвич-варианте (ELISA на твердой фазе иммобилизуют антитело, с которым связывается антиген, который является мишенью анализа, а затем с антигеном, связавшимся с первым антителом, взаимодействует второе антитело. В ELISA используются два типа антител, которые являются антителом или фрагментом, специфичным к антигену, который надо выявить или количественно определить, и имеющих разные антиген-связывающие домены: первый – это антитела или фрагменты антитела, которые заранее адсорбируют на планшете (например, 96-луночном); вторые антитела или фрагменты антитела предварительно метят флуоресцентным агентом, например FITC, ферментом, например пероксидазой, биотином и др. Взятые из организма клетки, или ткани, или их гомогенаты; супернатант культуры клеток; сыворотку крови; плевральный выпот; асцитную жидкость; внутриглазную жидкость и проч. наносят на планшет с иммобилизованными первыми антителами, затем прибавляют меченые антитела или фрагменты антител и происходит выявляющая реакция соответственно метке. Для количественного определения строят калибровочную кривую по данным анализа с серийными разведениями антигена известной концентрации, с помощью которой рассчитывают содержание антигена в образце.
В сэндвич-варианте ELISA можно использовать поликлональные антитела, моноклональные антитела и фрагменты антитела , например Fab, Fab’ или F(ab)2. В качестве двух типов антител в сэндвич-варианте ELISA можно сочетать моноклональные антитела или фрагменты антител, связывающиеся с разными эпитопами, либо сочетать поликлональные и моноклональные антитела или фрагменты антител .
Примером флуоресцентного ииммунологического анализа является метод, описанный в работах: Monoclonal Antibodies - Principles and Practice, 3-е издание, Academic Press (1996); Monoclonal Antibody Experimental Manual, Kodansha scientific books (1987) и др. В качестве метки используют известные флуоресцентные агенты [Fluorescent Antibody Method, Soft Science, Inc. (1983)], например FITC, RITC и т.п.
Примером люминесцентного иммунологического анализа является метод, описанный в работе Bioluminescence and Chemiluminescence Clinical Test 42, Hirokawa-Shoten Ltd. (1998) и в других подобных источниках. В таком анализе в качестве метки используют известные люминесцентные агенты, например акридиновый эфир, трифенилимидазол (лофин) и др.
Метод вестерн-блоттинга состоит в следующем: антиген, или клетку с антигеном, или иную субстанцию фракционируют путем электрофореза в полиакриламидном геле (PAGE) c додецилсульфатом натрия (SDS) [Antibodies - A Laboratory Manual Cold Spring Harbor Laboratory (1988)]. Затем с помощью блоттинга переносят молекулы из геля на поливинилиденфторидную (PVDF) или нитроцеллюлозную мембрану, дают мембране прореагировать с антителом или фрагментом антитела, связывающийся с искомым антигеном, и затем дают прореагировать с анти- IgG антителом или фрагментом данного антитела, меченного флуоресцентным агентом, например FITC, или ферментом, например пероксидазой, или биотином, или иным детектируемым агент, с последующей визуализацией метки. Пример такого анализа описан ниже.
Клетки или ткань, в которых экспрессируется полипептид с нужной аминокислотной последовательностью, подвергают лизису и лизат анализируют путем SDS-PAGE в восстанавливающих условиях, нанося аналит с рассчетом 0,1-30 мкг белка на одну дорожку. После электрофореза белок переносят на мембрану PVDF и блокируют сайты раствором PBS, содержащим 1-10% BSA (далее обозначается BSA-PBS) в течение 30 минут при комнатной температуре. Затем проводят реакцию с биспецифическими антителами по данному изобретению, после чего промывают мембрану PBS, содержащим 0,05 – 0,1% Tween 20 (Tween-PBS)), и проводят обработку козьими антителами к мышиному IgG, меченными пероксидазой, в течение 2 часов при комнатной температуре. Промывают мембрану Tween-PBS и выявляют полосу, с которой связались антитела, используя ECL Western Blotting Detection Reagents (производство Amersham, Inc.) или подобное средство детекции; таким образом выявляется искомый антиген. В вестерн-блоттинге используют антитела, способные связываться с полипептидом, не сохранившим природной конформации.
К числу физико-химических методовпринадлежат, например, такие, где детектируется агрегат, образованный при связывании по меньшей мере одного антигена, а именно, TfR или антигена клеточной поверхности, например EGFR, с биспецифическим антителом или биспецифическим фрагментом антитела . Еще одним примером физико-химического метода является метод капиллярной трубки, одномерная иммунодиффузия, иммунотурбидиметрический анализ, в том числе с латексными микрочастицами [Outline of Clinical Examination Method, KANEHARA & Co., LTD. (1998)] и другие подобные методы.
При латексной иммунотурбидиметрии частицы носителя, например полистирольного латекса, диаметром около 0,1-1 мкм сенсибилизируют, присоединяя к нему молекулы антитела (или антигена), так что образование комплекса антитело-антиген происходит на частицах, благодаря чему свет в реакционной смеси рассеивается сильнее, а проходит через нее меньше. Концентрацию антигена в образце определяют по изменению поглощения или рассеяния света, обусловленному взаимодействием со специфичным к нему антителом на частицах.
Для выявления или количественного определения клеток, в которых экспрессируются TfR , или антиген клеточной поверхности, например EGFR, или оба этих вещества, можно применять известные иммунологические методы, но предпочтительны иммунопреципитация, иммуноцитологическое или иммуногистохимическое окрашивание, иммунофлуоресцентный анализ и подобные им методы.
При иммунопреципитации клетка или иной агент, где экспрессируется TfR и/или антиген клеточной поверхности, например, EGFR, взаимодействует с биспецифическим антителом или биспецифическим фрагментом антитела по данному изобретению, после чего прибавляют носитель, обладающий способностью специфично связывать иммуноглобулины, например протеин G-сефарозу, в результате чего комплекс антиген-антитело осаждается.
В другом варианте метода биспецифическое антитело или биспецифический фрагмент антитела по данному изобретению иммобилизуют на 96-луночном планшете для ELISA и и блокируют планшет BSA-PBS. Затем BSA-PBS сливают, планшет промывают PBS и проводят реакцию с раствором лизата клеток или ткани, в которых экспрессируется TfR и/или антиген клеточной поверхности, например EGFR. После взаимодействия антигена с антителом планшет тщательно промывают и иммунопреципитат экстрагируют буферным раствором для образцов SDS-PAGE. Выявление или количественное определение проводят путем вестерн-блоттинга, описанного выше.
При иммуноцитологическом или иммуногистохимическом окрашивании клетки или ткань, в которых экспрессируется искомый антиген или подобное, обрабатывают поверхностно-активным веществом, или метанолом, или подобным агентом для увеличения проницаемости клеточной мембраны для антител, и проводят реакцию с биспецифическим антителом по данному изобретению, а затем анти-иммуноглобулиновое антитело или его связывающий фрагмент, меченые флуоресцентной меткой, например FITC или подобной, или ферментной, например пероксидазой, или биотином и т.п.; затем метку визуализируют и наблюдают с помощью микроскопа. Также выявление возможно путем иммунофлуоресцентного анализа, при котором с клетками взаимодействует флуоресцентно меченное антитело, а анализ проводят с помощью проточного цитометра [Monoclonal Antibodies - Principles and Practice, 3-е издание, Academic Press (1996), Monoclonal Antibody Experimental Manual, Kodansha scientific books (1987)]. В частности, биспецифические антитела или биспецифически фрагменты антител по данному изобретению позволяют выявлять TfR и/или антиген клеточной поверхности, например EGFR, экспрессирующиеся на клеточной мембране, путем иммунофлуоресцентного анализа.
Если для флуоресцентного мечения антител использовать систему FMAT 8100 HTS (производство Applied Biosystems, Inc.) или подобные ей, то можно измерить количество антигена или антитела без необходимости отделять комплекс антиген-антитело от свободных молекул антитела или антигена, не участвовавших в оюразовании комплекса антиген-антитело.
Примеры
Далее в настоящем документе данное изобретение описывается конкретно на примерах, которые, однако, не ограничивают объем изобретения.
Пример 1. Получение растворимых антигенов человеческих и обезьяньих TfR и EGFR
1. Получение растворимых антигенов
человеческого и обезьяньего TfR
Получали белки, являющиеся внеклеточными доменами человеческого и обезьяньего TfR, в которых к N-концу присоединен FLAG-Fс, как описано ниже. Нуклеотидная последовательность, кодирующая внеклеточный домен человеческого TfR, представлена SEQ ID NO: 1; выведенная по ней аминокислотная последовательность представлена SEQ ID NO: 2; нуклеотидная последовательность, кодирующая внеклеточный домен обезьяньего TfR, представлена SEQ ID NO: 3; выведенная по ней аминокислотная последовательность представлена SEQ ID NO: 4.
(1) Получение векторов для FLAG-Fc-человеческий TfR и FLAG-Fc-обезьяний TfR
Синтезировали фрагмент гена внеклеточного домена человеческого TfR, состоящий из нуклеотидной последовательности, представленной SEQ ID NO: 1, на основе нуклеотидной последовательности гена человеческого TfR (регистрационный номер в базе данных GenBank NM_003234.2, SEQ ID NO: 5; аминокислотная последовательность, кодируемая этой нуклеотидной последовательностью, представлена SEQ ID NO: 6).
Вектор INPEP4 (производство Biogen-IDEC GmbH), содержащий последовательноститэг FLAG и области Fc человеческого IgG, расщепляли рестриктазами BamHI и KpnI, и встраивали в вектор нуклеотидную последовательность, представленную SEQ ID NO: 1; таким образом получался экспрессионный вектор для образования FLAG-Fc-человеческий TfR.
Таким же путем получали экспрессионный вектор для образования FLAG-Fc-обезьяний TfR, содержащий фрагмент гена внеклеточного домена обезьяньего Tfr, состоящий из нуклеотидной последовательности, представленной SEQ ID NO: 3, на основе нуклеотидной последовательности гена обезьяньего TfR (SEQ ID NO: 7; аминокислотная последовательность, кодируемая этой нуклеотидной последовательностью, представлена SEQ ID NO: 8).
(2) Получение белков FLAG-Fc-человеческий TfR и FLAG-Fc-обезьяний TfR
Экспрессионный вектор, кодирующий FLAG-Fc-человеческий TfR, полученный в 1-(1), ввели в клетки линии эмбриональных почек человека HEK 293, используя экспрессионную систему FreeStyle (товарный знак) 293 (производство Thermo Fisher, Inc.) и культивировали клетки, чтобы произошла временная экспрессия указанного белка. Через 5 суток после введения вектора в клетки собирали супернатант клеточной культуры и фильтровали его через мембранный фильтр (производство Millipore Corporation) с порами диаметром 0,22 мкм.
Культуральный супернатант подвергали аффинной очистке на смоле с протеином А (MabSelect, производство GE Healthcare, Inc.). Антитела, адсорбировавшиеся на протеине А, промывали солевым раствором, забуференным фосфатом, по Дульбекко [жидкий, без кальция и магния; далее в настоящем документе обозначается D-PBS(-), производство Nacalai Tesque, Inc.], элюировали буферным раствором цитрата натрия (100 мМ; рН 3,5) и собирали в пробирку с раствором Tris-HCl (2М; pH 8,5).
Затем последний буферный раствор заменяли на D-PBS(-) путем ультрафильтрации, используя VIVASPIN (производство Sartоrius), после чего стерилизовали путем фильтрации, используя мембранный фильтр Millex-Gv (производство Millipore Corporation) с порами диаметром 0,22 мкм; в результате получали белок FLAG-Fc-человеческий TfR. Таким же образом получали белок FLAG-Fc-обезьяний TfR. используя экспрессионный вектор, кодирующий FLAG-Fc-обезьяний TfR, который получили по 1-(1). Определяли концентрацию полученных белков, измеряя поглощение на длине волны 280 нм и используя в расчетах коэффициент экстинкции, взятый на основании аминокислотной последовательности белка.
2. Получение растворимых антигенов человеческого и обезьяньего EGFR
Получали белки, являющиеся внеклеточными доменами человеческого и обезьяньего EGFR, в которых к С-концу присоединен FLAG-Fс GST как описано ниже. Нуклеотидная последовательность, кодирующая сигнальную аминокислотную последовательность и внеклеточный домен человеческого EGFR, представлена SEQ ID NO: 9; выведенная по ней аминокислотная последовательность представлена SEQ ID NO: 10. Нуклеотидная последовательность, кодирующая сигнальную аминокислотную последовательность и внеклеточный домен обезьяньего EGFR, представлена SEQ ID NO: 11; выведенная по ней аминокислотная последовательность представлена SEQ ID NO:12.
Синтезировали фрагмент гена внеклеточного домена человеческого EGFR, состоящий из нуклеотидной последовательности, представленной SEQ ID NO: 9, на основе нуклеотидной последовательности гена человеческого EGFR (регистрационный номер в базе данных GenBank NM_005228.4, SEQ ID NO: 13; аминокислотная последовательность, кодируемая этой нуклеотидной последовательностью, представлена SEQ ID NO: 14). Таким же образом синтезировали фрагмент гена внеклеточного домена обезьяньего EGFR, состоящий из нуклеотидной последовательности, представленной SEQ ID NO: 11. Белки человеческого EGFR-FLAG-Fc, обезьяньего EGFR-FLAG-Fc, человеческого EGFR- GST, обезьяньего EGFR- GST получали, как описано в пунктах 1-(1) и (2).
Определяли концентрацию полученных белков, измеряя поглощение на длине волны 280 нм и используя в расчетах коэффициент экстинкции, взятый на основании аминокислотной последовательности белка.
Пример 2. Получение клеток СНО для экспрессии
человеческого или обезьяньего TfR на клеточной мембране
Ген человеческого TfR, представленный SEQ ID NO: 5. клонировали в векторе pKANTEX (описан в патенте CША № 6423511) и таким образом получили экспрессионный вектор pKANTEX-человеческий TfR (полный) для экспрессии человеческого TfR на клеточной мембране.
Полученный экспрессионный вектор pKANTEX-человеческий TfR (полный) ввели в клетки CHO и культивировали эти клетки, чтобы происходила временная экспрессия соответствующего белка. После введения гена отдельные единичные клетки культивировали для получения колоний и таким образом получили клетки человеческий TfR/CHO.
Таким же образом клонировали ген обезьяньего TfR, представленный SEQ ID NO: 7, и получили клетки обезьяний TfR/CHO.
Клетки, окрашенные внутриклеточным флуоресцентным красителем 5(6)-карбоксифлуоресцеиндиацетата N-сукцинимидиловым эфиром (CFSE), получали следующим путем. Клетки человеческий TfR/CHO и обезьяний TfR/CHO обрабатывали 0,02% EDTA-PBS (Nacalai Tesque, Inc.), собирали и прибавляли CFSE (Sigma-Aldrich Co. LLC) до конечной концентрации 0,2 мкМ; затем давали прореагировать при комнатной температуре в течение 10 минут, после чего к клеткам прибавляли раствор D-PBS(-), содержащий10% эмбриональной бычьей сыворотки (Nacalai Tesque, Inc.) и клетки инкубировали при температуре 37°C в течение 10 минут. После реакции клетки центрифугировали и промывали 1%-ным раствором BSA-PBS (Nacalai Tesque, Inc.), содержащим 0,02% EDTA и 0,05% NaN3. Полученные окрашенные CFSE клетки замораживали в среде CELLBANKER 1 (Nippon Zenyaku Kogyo Co., Ltd.).
Пример 3. Иммунизация животных для продуцирования
человеческих антител
Чтобы получить моноклональные антитела к человеческому TfR и человеческому EGFR, животных, у которых могут образовываться человеческие антитела, иммунизировали FLAG-Fc-человеческий TfR или человеческий EGFR-FLAG-Fc, смешанным с адъювантиом, в общей сложности 4-5 раз. У иммунизированных животных брали сыворотку крови и активность связывания с человеческим TfR или с человеческим EGFR определяли путем ELISA для подтверждения роста титра антител.
Пример 4. Получение антител к TfR
1. Получение фаговой М13 библиотеки человеческих антител с легкой цепью А27 после иммунизации TfR
РНК экстрагировали из клеток селезенки или мононуклеарных клеток периферической крови (PBMC), полученных от иммунизированных животных, с помощью набора RNeasy Mini kit (производство QIAGEN, Inc.). Амплифицировали кДНК с помощью набора SMARTer RACE cDNA amplification kit (производство Clontech Laboratories, Inc.), а затем амплифицировали фрагмент гена VH путем ПЦР. Фрагмент гена VH и нуклеотидную последовательность A27, которая является последовательностью зародышевой линии человеческого антитела и состоит из нуклеотидной последовательности, представленной SEQ ID NO: 15, встраивали в фагмидный вектор pCANTAB 5E (производство Amersham Pharmacia, Inc.), которым трансформировали клетки E. coli TG1 (производство Lucigen Corporation), получая таким образом плазмиды.
Отметим, что нуклеотидная последовательность A27 кодирует VL человеческого антитела, состоящую из аминокислотной последовательности, представленной SEQ ID NO: 16; аминокислотные последовательности CDR 1, 2 и 3 этой VL (обозначаются LCDR 1, 2 и 3 соответственно) представлены SEQ ID NOS: 17, 18 и 19 соответственно.
Инфицируя фаг-помощником VCSM13, устойчивый к интерференции (производство Agilent Technologies, Inc.), полученными плазмидами получали М13-фаговую библиотеку человеческого антитела, образовавшегося в результате иммунизации TfR, в которой ген VL состоит из нуклеотидной последовательности A27 и которая включает библиотеку генов VH.
2. Получение моноклонального антитела к TfR
с легкой цепью A27
Используя М13-фаговую библиотеку человеческого антитела, образовавшегося в результате иммунизации TfR, с помощью фагового дисплея получали моноклональное антитело к TfR, содержащее VL, кодируемую нуклеотидной последовательностью А27.
(1) Пэннинг белка с иммобилизацией на стенках пробирки.
На поверхности пробирок MAXISORP STAR TUBE (производство NUNC, Inc.) иммобилизовали человеческий TfR (производство BBI Solutions, Inc.) и ту часть, с которой человеческий TfR не связался, блокировали реагентом SuperBlock Blockig Buffer (производство Thermo Fisher, Inc.); вносили М13-фаговую библиотеку человеческого антитела, реакция протекала при комнатной температуре в течение 1-2 часов; затем промывали 3 раза D-PBS(-) и PBS, содержащим 0,1% Tween 20 (далее в настоящем документе обозначается PBS-T; производство Wako Pure Chemical Industries, Ltd.), после чего связавшиеся фаговые частицы элюировали буферным раствором глицин-HCl (0,1 M; pH 2,2); элюат нейтрализовали буферным раствором Tris-HCl (2М; pH 8,5).
(2) Пэннинг клеток СНО, на мембране которых экспрессируется человеческий либо обезьяний TfR
М13-фаговую библиотеку человеческого антитела инкубировали с клетками CHO на льду в течение 30 минут; центрифугировали, чтобы отделить клетки и собрать супернатант, получая таким образом раствор с фаговыми частицами, взаимодействовавшими с клетками СНО. Затем раствор фагов инкубировали с человеческим TfR/CHO, окрашенными CFSE, которые получили по Примеру 1, на льду от 30 минут до 1 часа, после чего центрифугировали и супернатант отбрасывали. Промывали несколько раз раствором D-PBS(-, содержащим 5% эмбриональной бычьей сыворотки, 1 мM EDTA и 0,1% NaN3 (далее в настоящем документе называется буферным раствором SM), после чего прибавляли первичные антитела – антитела к M13 (GE Healthcare, Inc.) и позволяли прореагировать выдерживая на льду в течение 30 минут. Центрифугировали, супернатант отбрасывали, осадок промывали несколько раз буферным раствором SM, после чего прибавляли вторичные антитела – козьи антитела к мышиному IgG (H+L), меченные аллофикоцианином (APC; Southern Biotech, Inc.) и позволяли прореагировать выдерживая на льду в течение 30 минут. Центрифугировали, супернатант отбрасывали, осадок промывали несколько раз буферным раствором SM; затем прибавляли 7-aминоактиномицин D (AAD; BD Biosciences, Inc.) и позволяли прореагировать выдерживая на льду на 30 минут. При помощи проточного цитометра (BD Biosciences, Inc., FACS Aria III) отбирали жизнеспособные клетки, не окрашивающиеся 7-ADD и несущие антиген, окрашивающиеся CFSE. Кроме того, когда подтверждалось связывание фаговых частиц с клетками, в которых экспрессировался данный антиген, отбирали также клетки, связавшие фаг и окрашивающиеся APC. Связавшиеся с отобранными клетками фаговые частицы элюировали буферным раствором глицин-HCl (0,1 М; pH 2,2); элюат нейтрализовали буферным раствором Tris-HCl (2 М; pH 8,5).
Пэннинг клеток где использовались клетки обезьяний TfR/СНО проводиди аналогичным образом. Элюированным фагом заражали компетентные клетки E. coli TG1 для его амплификации. Действия, описанные выше в (1) или (2) повторяли 2-3 раза, чтобы сконцентрировать фаг, экспонирующий scFv, которые специфично связываются с человеческим TfR. Концентрированный фаг использовали для заражения клеток E. coli TG1, которые после этого высевали на чашки со средой SOBAG (2,0% триптон; 0,5% дрожжевой экстракт; 0,05% NaCl; 2,0% глюкоза; 10 мM MgCl2; 100 мкг/мл ампициллин; 1,5% агар), чтобы образовались колонии.
Колонии переносили на свежую среду, культивировали и заражали фагом-помощником, устойчивым к интерференции, VCSM13 и снова культивировали; таким образом получался моноклональный фаг. Используя полученный моноклональный фаг, с помощью проточного цитометра (Millipore Corporation, Guava easy Cyte HT) отбирали клон, связывающийся как с человеческим, так и с обезьяньим TfR .
При проточной цитометрии фаговый клон добавляли к клеткам человеческий TfR/CHO либо обезьяний TfR/CHO по Примеру 2. Инкубировали при температуре 4°C в течение 30 минут для проведения реакции, после чего каждую лунку промывали 3 раза раствором PBS-T.
Для оценки активности связывания моноклонального фага с TfR проводили ELISA. А именно, раствор с фагом, разбавленный PBS-T, содержащим 10% блокирующего реагента Block Ace (производство Dainippon Pharmaceutical Co., Ltd.), вносили в лунки 96-луночного иммунологического планшета (NUNC, Inc.) с иммобилизованным антигенным белком, инкубировали при комнатной температуре в течение 1 часа для проведения реакции, после чего промывали раствором PBS-T и добавляли меченные пероксидазой хрена антитела к M13 (производство GE Healthcare, Inc.), разведенные в 1%-ном растворе BSA-PBS, и инкубировали при комнатной температуре в течение 1 часа для проведения реакции. Затем планшет промывали PBS-T, прибавляли раствор хромогенного субстрата TMB (производство DAKO, Inc.) и оставляли при комнатной температуре для протекания цветной реакции., которую останавливали добавлением соляной кислоты (2 н); измеряли поглощение на длине волны 450 нм (референсная длина волны 570 нм) с помощью планшетного ридера (Emax, Molecular Devices, Inc.).
Анализировали последовательности для клонов, связывавшихся как с человеческим, так и с обезьяньим TfR; таким образом получали антитела к TfR, в которых VL кодировалась нуклеотидной последовательностью А27. В таблице 1 приведены номера (SEQ ID NO) полных нуклеотидных последовательностей, кодирующих VH каждого из полученных антител к TfR, выведенные по ним аминокислотных последовательностей и аминокислотных последовательностей CDR 1-3 VH (далее иногда также обозначаются HCDR 1-3). Значок “-” означает, что для данной последовательности нет информации.
Таблица 1. Номера последовательностей вариабельной области
тяжелой цепи (VH) антител к TfR
последовательность VH
3. Получение антител к TfR,
в которых легкая цепь отлична от A27
(1) Получение гибридом слиянием клеток
Мышиные миеломные клетки линии P3-U1 (P3X63Ag8U.1, регистрационный номер в АТСС CRL-1597) культивировали в среде S-Clone (EIDIA Co., Ltd.), адаптируя к бессывороточным условиям, а затем использовали в качестве родительской линии для клеточного слияния. У иммунизированных животных в асептических условиях извлекали селезенку , проводили гемолиз, используя RED BLOOD CELL LYSING BUFFER (Sigma-Aldrich Co. LLC); полученные клетки дважды промывали PBS и смешивали с клетками P3-U1 в соотношении (селезеночные клетки) : (P3-U1) = 8:1; клеточную смесь центрифугировали со скоростью 1200 об/мин в течение 5 минут.
Полученный тщательно разрыхляли клеточные кластеры в образовавшемя осадке, и добавляли 0,5 мл смешанного раствора 1 г полиэтиленгликоля-1000 (PEG-1000, Junsei Chemical Co., Ltd.), 1 мл среды MEM (Nacalai Tesque, Inc.) и 0,35 мл диметилсульфоксида (Sigma-Aldrich Co. LLC) при температуре 37°C, затем прибавляли среду МЕМ – сначала по 1 мл 5 раз с интервалом 1 мин, затем до общего объема 50 мл.
Суспензию клеток центрифугировали со скоростью 900 об/мин в течение 5 минут, и клетки в полученном осадке осторожно разрыхляли и суспендировали клетки селезенки при плотности 1,5 × 107 клеток/9 мл в среде S-Clone, содержащей HAT SUPPLEMENT; (Thermo Fisher Scientific, Inc.). В лунки 96-луночного планшета заранее вносили по 100 мкл среды S-Clone с добавкой HAT, в подготовленные таким образом лунки вносили по 100 мкл клеточной суспензии и культивировали в CO2-инкубаторе (5% CO2) при температуре 37°С в течение 8-10 суток.
(2) Скрининг гибридом
Определяли активность связывания TfR антителами, содержащимися в супернатанте культуры гибридом, методом ELISA. Образцы брали из супернатанта культуры гибридом, измерения проводили, как описано выше в разделе 2.(2).
(3) Субклонирование гибридом
Клетки из лунок, положительных по результатам скрининга, субклонировали и культивировали в среде для клонирования S-Clone в течение 7-10 суток.
(4) Клонирование и секвенирование гена вариабельной области антитела
Из клонированных гибридом выделяли суммарную РНК, используя набор RNeasy Mini kit (QIAGEN, Inc.) и амплифицировали гены антител методом 5’-RACE. Для синтеза кДНК для RACE использовали набор SMARTer RACE Kit (Clontech Laboratories, Inc.). Амплифицированную последовательностьвариабельной области антитела, клонировали с помощью набора TOPO TA Cloning (Invitrogen, Inc.), чтобы подтвердить последовательность нужного фрагмента ДНК.
Из числа полученных антителах к TfR аминокислотные последовательности, выведенные по нуклеотидным последовательностям, кодирующим вариабельные области тяжелой и легкой цепей клона 2230, представлены SEQ ID NO: 53 и SEQ ID NO: 54 соответственно.
4. Конструирование вектора для экспрессии
полученных антител к TfR
Получали векторы для экспрессии растворимых IgG со встроенными в них генами полученных антител к TfR. Вначале ген A27, кодирующий общую VL, субклонировали в векторе N5KG4PE (IDEC Pharmaceuticals) или N5KG4PE R409K (описан в публикации WO 2006/033386).
Затем ген VH субклонировали в N5KG4PE или N5KG4PE R409K; таким образом получались экспрессионные векторы для получения моноклональных антител к TfR с константной областью человеческого IgG4PE или с константной областью человеческого IgG4PE R409K.
Чтобы использовать в качестве положительного контроля моноклональные антитела к TfR TfR434 и TfR435, которые являются нейтрализующими TfR антителами (описаны в публикации WO 2012/153707), получали соответствующие экспрессионные векторы. Аминокислотные последовательности VH антител TfR434 и TfR435 представлены SEQ ID NOS: 55 и 56 соответственно, аминокислотная последовательность VL представлена SEQ ID NO: 57.
Синтезировали гены, кодирующие VH и VL антитела TfR435, и субклонировали их в векторе N5KG1 (описан в публикации WO 2003/033538); таким образом получался экспрессионный вектор для моноклонального антитела к TfR TfR435 с константной областью человеческого IgG1.
Далее в настоящем документе все эти моноклональные антитела называются обозначениями соответствующих клонов.
Пример 5. Получение антител к антигену клеточной поверхности
I. Получение антител к EGFR
1. 1. Получение М13-фаговой библиотеки человеческого антитела после иммунизации EGFR
М13-фаговую библиотеку человеческого антитела после иммунизации EGFR с геном VL, состоящим из нуклеотидной последовательности A27, или с геном VL антитела к TfR 2230, включающую библиотеку генов VH, получали так же, как описано в Примере 4, 1.
2. Получение антител к EGFR
На пробирках MAXISORP STARTUBE (производство NUNC, Inc.) иммобилизовали человеческий или же обезьяний белок EGFR-Fc, полученный по Примеру 1, и ту часть, с которой не связался антиген, блокировали реагентом SuperBlock Blockig Buffer (производство Thermo Fisher, Inc.); после иммунизации EGFR получали библиотеку человеческих антител с фагом М13, экспонирующим scFv, которые специфично связываются с человеческим EGFR и обезьяньим EGFR, и моноклонировали так, как описано в Примере 4, 2.
Указанные действия повторяли 3-5 раз, чтобы сконцентрировать фаг, экспонирующий scFv, специфично связывающиеся с человеческим EGFR-Fc и обезьяньим EGFR-Fc.
Потверждение, что клон специфически распознает человеческий EGFR-Fc и обезьяний EGFR-Fc проводили методом ELISA, который проводили, как описано в Примере 4, с иммобилизацией человеческого EGFR-Fc или обезьяньего EGFR-Fc по Примеру 1.
Из числа клонов, которые связывались как с человеческим, так и с обезьяньим EGFR, отобрали три клона с высокой аффинностью связывания, сравнимым с таковым цетуксимаба. Для всех этих трех клонов провели анализ последовательностей и получили антитела к EGFR с VL, кодируемой нуклеотидной последовательностью A27. Сведения о VH этих антител к EGFR представлены в таблице 2 по той же схеме, что и в таблице 1.
Таблица 2. Последовательности VH антител к EGFR
кодирующая VH
HCDR1
HCDR2
HCDR3
Аналогично были получены антитела к EGFR с такой же VL, как у антител к TfR 2230. Из числа полученных антител к ЕGFR для клонов KME07, KME09 и KME11 по нуклеотидным последовательностям, кодирующим вариабельные области тяжелых цепей, были выведены аминокислотные последовательности, которые представлены SEQ ID NO: 73, SEQ ID NO: 74 и SEQ ID NO: 75 соответственно.
Были получены векторы для экспрессии растворимых IgG со встроенными в них генами, кодирующими антитела к ЕGFR, так же, как описано в Примере 4. В качестве константной области использовали константную область человеческих антител IgG4PE или IgG4PE R409K.
В качестве положительного контроля для антител к EGFR получали антитело с вариабельной областью моноклонального антитела С225 к EGFR, описанного в публикации WO 1996/040210 (далее в настоящем документе оно называется цетуксимабом). Аминокислотная последовательность VH цетуксимаба представлена SEQ ID NO: 76. Аминокислотная последовательность VL цетуксимаба представлена SEQ ID NO: 77.
Синтезировали гены, кодирующие VH и VL цетуксимаба, и субклонировали их в векторе N5KG1 так же, как описано в Примере 4; таким образом получили экспрессионный вектор для цетуксимаба с константной областью человеческого IgG1.
II. Получение антител против Глипикана-3
Получали антитело с вариабельной областью анти-глипикан-3 моноклонального антитела HN3, описанного в публикации WO 2012/145469. Аминокислотная последовательность VH антитела HN3 представлена SEQ ID NO: 78. Синтезировали ген, кодирующий VH HN3, и субклонировали его в векторе N5KG4PE R409K, как описано в Примере 4; таким образом получали анти-глипикан-3 моноклональное антитело HN3 с константной областью человеческого IgG4PE R409K.
Пример 6. Конструирование экспрессионного вектора для получения биспецифического антитела, связывающегося с TfR
и с антигеном клеточной поверхности
Биспецифическое антитело, имеющее структуру, изображенную на фиг. 1(A), и связывающееся с человеческим и с обезьяньим TfR, а также с человеческим и с обезьяньим EGFR, получали следующим образом. На фиг. 1(A) - 1(C) в изображенных антителах VH в составе полипептида N-концевой стороны, обозначена VH1, а VH в состве части IgG С-концевой стороны, обозначена VH2. Эти биспецифические антитела имеют форму, описанную в публикации WO 20 9/131239.
Области VH1 и VH2 – это VH антитела, полученного в Примере 4 либо 5, и VH1 и VH2 являются VH антител против разных антигенов.
Далее в настоящем описании в тех случаях, когда полипептид N-концевой стороны и антитело изотипа IgG соединены посредством линкера, ген, кодирующий аминокислотную последовательность линкера, называется геном линкера.
В биспецифических антителах, получаемых, как описано ниже, в качестве линкера использовалась аминокислотная последовательность с частью шарнирного участка IgG4 [ES (SEQ ID NO: 79), ESKYG (SEQ ID NO: 80), ESKYGPP (SEQ ID NO: 81) и др.], или линкер типа GS (GGGGS (SEQ ID NO: 82)], или последовательность из трех повторов GGGGS). В качестве константной области тяжелой цепи части IgG использовалась константная область тяжелой цепи IgG4PE или IgG4PE R409K (кодирующая ее нуклеотидная последовательность представлена SEQ ID NO: 83, соответствующая аминокислотная последовательность представлена SEQ ID NO: 84), описанные в публикации WO 2006/033386.
Далее в настоящем документе биспецифическое антитело, в котором полипептид N-концевой стороны включает вариабельную область антитела к EGFR и часть IgG включает вариабельную область антитела к TfR, называется EGFR-TfR-биспецифическим антителом. Также, например, EGFR-TfR -биспецифическое антитело, включающее вариабельную область антитела Е12 к EGFR и вариабельную область антитела TfR1071 к TfR, называется E12-TfR1071-биспецифическим антителом. Аналогично, биспецифическое антитело, в котором полипептид N-концевой стороны включает вариабельную область антитела к TfR и часть IgG включает вариабельную область антитела к EGFR, называется TfR-EGFR-биспецифическим антителом.
Также, например, TfR-EGFR-биспецифическое антитело, включающее вариабельную область антитела TfR1071 к TfR и вариабельную область антитела E12 к EGFR называется TfR1071-E12-биспецифическим антителом. Антитело, в котором между полипептидом N-концевой стороны и частью IgG имеется линкер, называется EGFR-линкер-TfR-биспецифическим антителом.
Также, например, EGFR-линкер-TfR-биспецифическое антитело, включающее вариабельную область антитела Е08 к EGFR, вариабельную область антитела TfR1071 к TfR и линкер ES, называется E08-ES-TfR1071-биспецифическим антителом. Заметим, что если линкером служит GS (SEQ ID NO: 82), то в названии биспецифического антитела он обозначается GS, а если линкером служит последовательность, в которой линкер GS повторяется 3 раза, то в названии биспецифического антитела он обозначается GS3.
Обозначения, структура и линкеры биспецифических антител по данному изобретению, обозначения клонов антител к TfR, использовавшихся для получения биспецифических антител, и обозначения клонов антител к антигену клеточной поверхности приведены в таблице 3. Отметим, что в этой таблице знак «-« означает, что линкер отсутствует, и соответствующая последовательность не приводится.
Таблица 3
биспецифического антитела
1. Получение экспрессионного вектора для биспецифического антитела
с легкой цепью общего типа
Экспрессионный вектор для биспецифического антитела с легкой цепью общего типа, изображенного на фиг. 1(A), из числа биспецифических антител, представленных в таблице 3, получали описанным ниже способом.
Вначале нуклеотидную последовательность A27, кодирующую общую VL субклонировали в векторе N5KG4PE R409K (описанном в публикации WO 2006/033386).
Затем, взяв в качестве матрицы ген (SEQ ID NO: 85), кодирующий такую же аминокислотную последовательность, какую имеет область CH1 человеческого IgG4, с измененными кодонами, амплифицировали данный фрагмент гена путем ПЦР, используя реагент KOD-Plus-Ver.2 (Toyobo Co., Ltd.). Используя в качестве матрицы ген VH антитела, полученного в примере 4 или 5, таким же образом амплифицировали фрагмент нуклеотидной последовательности, кодирующей VH2.
Полученные два фрагмента субклонировали в экспрессионном векторе для антитела по Примеру 4 или 5 между последовательностями, кодирующими VH1 и CH1 части IgG; таким образом получался экспрессионный вектор для нужного биспецифического антитела.
2. Получение экспрессионного вектора для биспецифического антитела
с легкой цепью не общего типа
Экспрессионный вектор для биспецифического антитела с легкой цепью не общего типа, изображенного на фиг. 1(A) из числа биспецифических антител, представленных в таблице 3, получали описанным ниже способом.
Антитело, изображенное на фиг. 1(B), образуется с помощью экспрессионного вектора определенного типа. Вначале нуклеотидную последовательность A27, кодирующую VL, субклонировали в векторе pCI-OtCAG-G4PE R409, содержащем нуклеотидную последовательность, кодирующую константную область человеческого IgG4PE R409K.
Этот вектор обозначается pCI-A27. Константная область, кодируемая нуклеотидной последовательностью в составе вектора pCI-OtCAG_hG4PE R409K, является константной областью тяжелой цепи мутантного IgG4 [далее в настоящем документе обозначается IgG4PE R409K (WO 2006/033386)], полученного в результате замены Ser в положении 228 на Pro, Leu в положении 235 на Glu и Arg в положении 409 на Lys (нумерация по индексу EU) в аминокислотной последовательности константной области тяжелой цепи человеческого IgG4.
Отметим, что такой вектор получают путем полного синтеза с введением сайтов рестрикции, необходимых для экспрессии гена человеческого антитела с использованием вектора pCI (Promega Corporation) в качестве основы.
После того, как встроены нуклеотидные последовательности, кодирующие VH и линкер антитела по Примеру 5, полностью синтезированную нуклеотидную последовательность, в которой лигированы нуклеотидная последовательность, кодирующая линкер, и ген VH антитела к TfR по Примеру 4, субклонировали в векторе pCI-A27; таким образом получался экспрессионный вектор для нужного антитела.
Пример 7. Получение моноклонального антитела, связывающегося
с TfR, моноклонального антитела, связывающегося
с антигеном клеточной поверхности, и биспецифического антитела
Проводили экспрессию и очистку моноклонального антитела, связывающегося с TfR, моноклонального антитела, связывающегося с антигеном клеточной поверхности, и биспецифического антитела, содержащего cвязывающие домены к обоим антигенам, полученных с использование экспрессионных векторов для антител по Примерам 4-6, следующим образом.
Вектором для экспрессии антитела ко-трансфицирповали клетки линии Expi293, используя набор Expi293 (торговое название) Expression System (Thermo Fisher, Inc.), спустя 12-16 часов прибавляли усилитель трансфекции; таким образом обеспечивалась временная экспрессия антитела или биспецифического.
Через 4-7 дней после введения вектора собирали культуральный супернатант, и фильтровали через мембранный фильтр (производство Millipore Corporation) с порами диаметром 0,22 мкм, после чего проводили аффинную очистку антитела, используя смолу с протеином А (MabSelect SuRe, GE Healthcare, Inc.). Промывали раствором D-PBS(-). Искомое антитело, адсорбированное на смоле с протеином А, элюировали буферным раствором цитрата натрия (100 мМ; pH 3,5) и собирали в пробирки, содержащие буферный раствор Tris-HCl (2 M; pH 8,5).
Полученный белковый раствор концентрировали при помощи VIVASPIN (Sartоrius), заменяли буферный раствор на D-PBS(-), используя колонку Nap (производство GE Healthcare, Inc.). В том случае. когда очищаемый компонент был мономерным, эту мономерную фракцию выделяли из раствора антител или биспецифических антител с помощью хроматографической системы AKTA FPLC (производство GE Healthcare, Inc.) и колонки для высокоэффективной очистки Superdex (производство GE Healthcare, Inc.). Затем стерилизовали путем фильтрации в системе AKTA (Millex-Gv, производство Millipore Corporation) на мембранном фильтре с порами диаметром 0,22 мкм; таким образом получали очищенное антитело или очищенное биспецифическое антитело.
В полученном таким образом растворе очищенного антитела или очищенного биспецифического антитела измеряли поглощение на длине волны 280 нм и определяли концентрацию очищенного антитела, используя коэффициент экстинкции, рассчитанный на основании аминокислотной последовательности антитела или биспецифического антитела.
Пример 8. Определение аффинности связывания биспецифических
антител с aнтигеном с помощью биосенсорной системы Biacore
Для определения активности связывания и межвидовой перекрестной реактивности EGFR-TfR-биспецифических антител, полученных по Примеру 7, к каждому из человеческого TfR, обезьяньего TfR, человеческого EGFR, обезьяньего EGFR использовали полученные по Примеру 1 белки FLAG-Fc-человеческий TfR, FLAG-Fc-обезьяний TfR, человеческий EGFR-GST, обезьяний EGFR-GST и определяли аффинность методом SPR с помощью оптической биосенсорной системы Biacore T100 (производство GE Healthcare, Inc.).
Антитела к человеческому IgG иммобилизовали на сенсорном чипе CM5 (производство GE Healthcare, Inc.), используя набор Human Antibody Capture Kit (производство GE Healthcare, Inc.) согласно прилагающейся инструкции. В проточную ячейку вносили испытуемое биспецифическое антитело в концентрации 1-3 мкг/мл на 10 секунд при скорости потока 10 мкл/мин.
Затем к качестве аналита вносили раствор каждого антигенного белка в HBS-EP+ (производство GE Healthcare, Inc.), содержащем 0,1% BSA в пяти трехкратных серийных разведениях от 1 нМ до 9 нМ – при скорости потока 30 мкл/мин и регистрировали ассоциацию биспецифического антитела и аналита в течение 2 минут, диссоциацию – в течение 10 минут.
Измерения проводились в режиме одноцикловой кинетики,. Сенсограммы анализировали с помощью программного обеспечения Bia Evaluation Software (производство GE Healthcare, Inc.) и для каждого антитела рассчитывали константу скорости реакции.
Рассчитанные значения константы диссоциации [kd/ka = KD] каждого из биспецифических антител при взаимодействии с человеческими и с обезьяньими TfR и и EGFR, а также значения отношения этой константы для связывания с человеческим белком к значению для связывания с обезьяньим белком приведены в таблице 4. Обозначение N/A указывает, что измерения в данном случае не проводились.
Таблица 4
(нМ)
обезьяний EGFR (нМ)
(нМ)
обезьяний TfR (нМ)
Как видно из таблицы 4, значения KD для связывания EGFR-TfR-биспецифических антител с человеческим и с обезьяньим TfR имеют порядок 10-8-10-9 M. Значения KD для связывания EGFR-TfR-биспецифических антител с обезьяньим TfR в 0,1-10 раз превышают значения KD для связывания EGFR-TfR-биспецифических антител с человеческим TfR; следовательно, указанные антитела обладают перекрестной реактивностью в отношении человеческого TfR и обезьяньего TfR.
Аналогично, значения KD для связывания EGFR-TfR-биспецифических антител с человеческим и обезьяньим рецепторами EGFR имеют порядок 10-9-10-10 M. Значения KD для связывания EGFR-TfR-биспецифических антител с обезьяним EGFR в 0,1-10 раз превышают значения KD для связывания EGFR-TfR-биспецифических антител с человеческим EGFR; следовательно, указанные антитела обладают перекрестной реактивностью в отношении человеческого EGFR и обезьяньего EGFR .
Пример 9. Определение уровня экспрессии антигена в раковых клетках методом проточной цитометрии и оценка аффинности связывания полученных антител
Уровень экспрессии различных антигенов в раковых клетках различных линий определяли методом сортировки клеток с активированной флуоресценцией (FACS) следующим образом. В способе использовали антитела к человеческого CD71 , меченные фикоэритрином (РЕ; BD Pharmingen, Inc.) и антитела к EGFR, меченные флуоресцентным красителем Alexa Fluor 488 (Santa Cruz Biotechnology, Inc.).
Определение уровня экспрессии TfR и EGFR в раковых клетках различных линий проводили следующим образом.
Клетки OE21 суспендировали в буферном растворе для окрашивания (SB), который представляет собой раствор D-PBS(-, содержащий 0,1% NaN3 и 1% FBS, до плотности 1 × 106 клеток в 1 мл; эту суспензию вносили по 100 мкл в лунки 96-луночного круглодонного планшета (производство Falcon, Inc.). Центрифугировали со скоростью 2000 об/мин в течение 2 минут при температуре 4°C, супернатант отбрасывали, а; к полученному осадку прибавляли антитела к человеческому CD71, меченные PE, или антитела к EGFR, меченные Alexa Fluor 488, и инкубировали в течение 30 минут в ледяной бане. Промывали клетки 2 раза раствором SB, суспендировали в этом же растворе и измеряли интенсивность флуоресценции в каждой лунке с помощью цитометра FACSCANTO II (производство BD Biosciences, Inc.). Так же определяли уровень экспрессии TfR в клетках линий T.Tn, U-937, HepG2, HuH-7 и HLE.
Аналогично определяли уровень экспрессии GPC3 в клетках HepG2, HuH-7 и HLE. А именно, клетки подготавливали, как описано выше, в качестве первичных антител использовали антитела HN3; в качестве вторичных антител - козьи антитела к человеческоиму IgG (H+L), меченные Alexa Fluor (зарегистрированный товарный знак) 488 (Molecular Probes, Inc.), затем инкубировали в течение 30 минут на ледяной бане; измеряли интенсивность флуоресценции, как описано выше.
В качестве отрицательного контроля использовали PBS и мутантные антитела IgG4 R409K (далее в настоящем документе называются антитела к DNP) полученные способом, описанным в Примере 5 публикации WO 2006/033386, с использованием вектора, кодирующего VH и VL (регистрационные номера в базе данных GenBank U16688 и U116687 соответственно) антитела к DNP-1, описанного в работе Mol. Immunol. 1996 Jun.; 33 (9): 759-68.
На фиг. 2(A)-2(C) изображены результаты определения методом проточной цитометрии уровня экспрессии EGFR в клетках OE21, T.Tn и U-937 соответственно. На фиг. 2(D)-2(F) изображены результаты определения методом проточной цитометрии уровня экспрессии TfR в клетках OE21, T.Tn и U-937 соответственно. Как видно из этих иллюстраций, в клетках OE21 уровень экспрессии EGFR высокий, в клетках T.Tn средний, в клетках U-937 низкий.
На фиг. 3(A)-3(C) изображены результаты определения методом проточной цитометрии уровня экспрессии GPC3 в клетках HepG2, HuH-7 и HLE соответственно. На фиг. 3(D)-3(F) изображены результаты определения методом проточной цитометрии уровня экспрессии TfR в клетках HepG2, HuH-7 и HLE соответственно. Как видно из этих иллюстраций, уровень экспрессии GPC3 в клетках HepG2 высокий, в клетках HuH-7 средний, а в клетках HLE этот белок не экспрессируется.. Во всех клетках: HepG2, HuH-7 и HLE – уровень экспрессии EGFR низкий.
Судя по фиг. 2 и 3, в исследованных линиях раковых клеток экспрессия TfR различалась незначительно, причем уровень экспресиии этого белка был от низкого до среднего.
Методом проточной цитометрии была подтверждена аффинность связывания антител к TfR и антител к EGFR, полученных по Примерам 4 и 5, к клеткам OE21. На фиг. 4(A)-4(I) изображены результаты, полученные с антителами E08, E12, E17, KME07, KME09, KME11, TfR1071, 2230 и T14 соответственно.
Из всех антител к EGFR связывание с клетками OE21 не подтвердилось только в случае антител KME11. Антитело KME11 связывалось с растворимым белком EGFR, описанным в Примере 5, но не связывалось с EGFR, ассоциированным с мембраной на клеточной поверхности. Из всех антител к TfR связывание с клетками OE21 не подтвердилось только в случае антител T14. Антитело T14 связывалось с растворимым TfR (по данным ELISA, см. Пример 4), но не связывалось с TfR, ассоциированным с мембраной на клеточной поверхности.
Пример 10. Определение подавления клеточного роста антителами к TfR
Способность моноклональных антител к TfR, полученных в Примере 4, подавлять клеточный рост, определяли, как описано ниже, взяв за показатель подавление пролиферации раковых клеток определенной линии.
Клетки линии OE21 высевали (по 1 × 103 клеток) в лунки плоскодонного 96-луночного планшета (производство Falcon, Inc.) и добавляли испытываемое антитело, разведенное в среде RPMI 1640 (производство Sigma-Aldrich Co. LLC), содержащей 10% FBS, до конечной концентрации 1 мкг/мл; инкубировали при температуре 37°C в атмосфере, содержащей 5,0% углекислого газа, в течение 4-6 суток. После этого использовали набор для люминесцентного анализа жизнеспособности клеток CellTiter-Glo (зарегистрированное торговое название) Luminescent Cell Vialbility Assay (производство Promega Corporation) и измеряли интенсивность флуоресценции с помощью ридера для микропланшетов (1420 ARVO Multi-Label Counter, производство WALLAC, Inc.). Для каждого варианта условий отводили по 6 лунок и рассчитывали среднее значение. Полученная для данной лунки интенсивность флуоресценции отражает уровень ATP в жизнеспособных клетках в этой лунке. В качестве отрицательного контроля использовали антитела к DNP.
Также вместе с испытываемым антителом добавляли поликлональные антитела к человеческоиму IgG (Sigma, Inc.) (далее в настоящем документе они называются перекрестно-сшивающими антителами) в концентрации 10 мкг/мл и в условиях, когда испытываемое антитело оказывается перекрестно-сшитым, проводили описанные выше измерения. Полученные данные представлены на фиг.5. Из числа испытываемых антител те моноклональные антитела, которые сами по себе не проявляли способность подавлять клеточный рост, отбирали как антитела, не способные нейтрализовать TfR .
При добавлении перекрестно-сшивающего антитела для перекрестной сшивки двух испытываемых антител, были выявлены те клоны, которые проявляли способность подавлять клеточный рост (TfR1071, TfR4016, cyno186, cyno292 и 2230) и те, которые не обладали этой способностью (PSM4072, TfR1007 и cyno163).
Пример 11. Определение подавления клеточного роста антителами
к антигену клеточной поверхности
Способность моноклональных антител к антигену клеточной поверхности, полученных в Примере 5, подавлять клеточный рост, определяли, как описано ниже, взяв за показатель подавление пролиферации раковых клеток определенной линии.
Клетки линии OE21 высевали (по 1 × 103 клеток) в лунки плоскодонного 96-луночного планшета (производство Falcon, Inc.) и добавляли испытываемое антитело, разведенное в среде RPMI 1640 (производство Sigma-Aldrich Co. LLC), содержащей 10% FBS, до различных концентраций; клетки культивировали при температуре 37°C в атмосфере, содержащей 5,0% углекислого газа, в течение 4-6 суток. После этого использовали набор для люминесцентного анализа жизнеспособности клеток CellTiter-Glo (зарегистрированное торговое название) Luminescent Cell Vialbility Assay (производство Promega Corporation) и измеряли интенсивность флуоресценции с помощью ридера для микропланшетов (1420 ARVO Multi-Label Counter, производство WALLAC, Inc.). Для каждого варианта условий отводили по 3 лунки и рассчитывали среднее значение. Полученная для данной лунки интенсивность флуоресценции отражает уровень ATP в жизнеспособных клетках в этой лунке В качестве отрицательного контроля использовали антитела к DNP.
Для определения подавления клеточного роста антителами к EGFR использовали клетки OE21. Полученные с этими клетками результаты представлены на фиг. 6. Как видно из фиг. 6, цетуксимаб и клон E08 имели высокую способность подавлять клеточный рост, антитела E17 проявляли эту способность слабо, а клон E12 ею вовсе не обладал. Эти результаты рассматриваются как отражение нейтрализующей активности указанных антител в отношении EGFR.
Пример 12. Определение подавления клеточного роста
биспецифическими антителами
Способность биспецифических антител, полученных в Примере 6, подавлять клеточный рост, определяли, как описано ниже, взяв за показатель подавление пролиферации раковых клеток определенной линии.
Раковые клетки высевали (по 1 × 103 клеток) в лунки плоскодонного 96-луночного планшета (производство Falcon, Inc.) и добавляли испытываемое антитело, разведенное в среде RPMI 1640 (производство Sigma-Aldrich Co. LLC), содержащей 10% FBS, до различных концентраций; клетки культивировали при температуре 37°C в атмосфере, содержащей 5,0% углекислого газа, в течение 4-6 суток. После этого использовали набор для люминесцентного анализа жизнеспособности клеток CellTiter-Glo (зарегистрированное торговое название) Luminescent Cell Vialbility Assay (производство Promega Corporation) и измеряли интенсивность флуоресценции с помощью ридера для микропланшетов (1420 ARVO Multi-Label Counter, производство WALLAC, Inc.). Для каждого варианта условий отводили по 3 лунки и рассчитывали среднее значение. Полученная для данной лунки интенсивность флуоресценции отражает уровень ATP в жизнеспособных клетках в этой лунке В качестве отрицательного контроля использовали антитела к DNP.
Для определения подавления клеточного роста биспецифическими антителами использовали клетки OE21. На фиг. 7А-7С представлены результаты, полученные для биспецифического антитела с вариабельной областью антитела E08 к EGFR, биспецифического антитела с вариабельной областью антитела E12 к EGFR и биспецифического антитела с вариабельной областью антитела к TfR 2230 соответственно.
Как видно из фиг. 7A, высокая способность подавлять клеточный рост наблюдалась в том случае, когда вариабельная область клона (например, TfR1071), проявлявшего способность подавлять клеточный рост в условиях перекрестной сшивки антительных молекул перекрестно-сшивающим антителом по Примеру 10, располагалась на стороне части IgG, а вариабельная область антитела к EGFR располагалась на стороне полипептида N-концевой стороны. Причем такая же способность подавлять клеточный рост наблюдалась, если в качестве линкера служил кусок аминокислотной последовательности шарнирного участка (ES, ESKYG или ESKYGPP) IgG4. А биспецифическое антитело, в котором имелась вариабельная область антитела к TfR в полипептиде N-концевой стороны, практически не подавляло клеточный рост.
Отметим, что, по результатам Примера 9, антитело Т14 к TfR T14 не взаимодействует с TfR на клеточной поверхности – следовательно, способность подавлять клеточный рост у антител E08-T14 и T14-E08 можно считать обусловленной подавлением сигнального пути с участием EGFR благодаря только E08 (Пример 11).
Как видно из фиг. 7B, биспецифические антитела, у которых в части IgG имеется вариабельная область какого-либо из клонов (R327, TfR1071, TfR4016, cyno186 и cyno292), способных подавлять клеточный рост в условиях перекрестной сшивки антительных молекул перекрестно-сшивающим антителом по Примеру 10, обладают большей способностью подавлять клеточный рост, нежели биспецифические антитела, у которых в части IgG имеется вариабельная область какого-либо из клонов (PSM4072, TfR1007 и cyno163) не способных подавлять клеточный рост в условиях перекрестной сшивки антительных молекул перекрестно-сшивающим антителом
Что касается биспецифических антител, содержащих вариабельную область антитела 2230 к TfR, то, как видно из фиг. 7C, антитела KME07-2230, KME09-2230 и KME11-2230 способны подавлять клеточный рост. Как указано в Примере 9, KME11 не связывается с EGFR на клетках - следовательно, биспецифическое антитело KME11-2230 обладает способностью подавлять клеточный рост не зависимым от EGFR образом. Есть основания опасаться, что клоны с таким свойством могут оказаться токсичными для нормальных клеток, у которых нет EGFR, но экспрессируется TfR , например для клеток костного мозга.
Описанные выше результаты показывают, что биспецифические антитела, содержащие в части IgG вариабельную область антител к TfR, которые сами по себе не способны подавлять клеточный рост, но проявляют эту способность будучи перекрестно-сшитыми (R327, TfR1071, TfR4016, cyno186 и cyno292), обладают высокой способностью подавлять клеточный рост избирательно к антигену клеточной поверхности. Кроме того, показано, что на способность биспецифических антител подавлять клеточный рост наличие или отсутствие линкера не влияет.
Чтобы доказать взаимосвязь между способностью EGFR-TfR-биспецифических антител подавлять клеточный рост и уровнем экспрессии EGFR, определяли указанную способность для биспецифических антител E12-TfR1071, полученных в Примере 6, в отношении различных линий клеток с разными уровнями экспрессии EGFR. Исходя из результатов Примера 9, в качестве клеток с высоким уровнем экспрессии EGFR использовали клетки OE21, со средним уровнем экспрессии EGFR - клетки T.Tn и с низким уровнем экспрессии EGFR - клетки U-93. Как видно из фиг.. 8A-8C, антитела E12-TfR1071 проявляли высокую способность подавлять пролиферацию клеток OE21, среднюю – в случае клеток T.Tn и не подавляли пролиферацию клеток U-937 Из этих результатов следует, что способность антитела E12-TfR1071 подавлять пролиферацию клеток зависит от уровня экспрессии в них EGFR.
Для определения подавления клеточного роста GPC3-TfR-биспецифическими антителами использовали клетки HepG2. Как видно из фиг. 9, антитела HN3-TfR1071, HN3-GS-TfR1071 и HN3-GS3-TfR1071 обладают очень высокой способностью подавлять клеточный рост.
Чтобы доказать взаимосвязь между способностью GPC3-TfR-биспецифических антител подавлять клеточный рост и уровнем экспрессии GPC3, определяли указанную способность для биспецифических антител HN3-TfR1071, полученных в Примере 6, в отношении различных линий клеток с разными уровнями экспрессии GPC3; подавление клеточного роста определяли так же, как в Примере 10.
Результаты, полученные с клетками HepG2, в которых уровень экспрессии GPC3 высокий, представлены на фиг. 10A; результаты, полученные с клетками HuH-7, в которых уровень экспрессии GPC3 средний, представлены на фиг. 10B; результаты, полученные с клетками HLE, в которых GPC3 не экспрессируется, представлены на фиг. 10C Как видно из фиг. 10A-10C, антитела HN3-TfR1071 значительно подавляли пролиферацию клеток HepG2, в которых уровень экспрессии GPC3 высокий, проявляли среднюю способность подавлять пролиферацию клеток HuH-7, в которых уровень экспрессии GPC3 средний, но не подавляли пролиферацию клеток HLE, в которых GPC3 не экспрессируется. Из этих данных следует, что антитело HN3-TfR1071 проявляет способность подавлять клеточный рост в зависимости от уровня экспрессии GPC3...
Также выяснилось, что в случае антигенов клеточной поверхности, отличных от EGFR, биспецифические антитела, содержащие вариабельную область антитела к TfR в части IgG, проявляют высокую способность подавлять клеточный рост, зависящую от антигена клеточной поверхности.
Пример 13. Подавление связывания трансферрина с TfR
антителами к TfR и биспецифическими антителами
к TfR
Чтобы установить механизм подавления роста раковых клеток биспецифическими антителами, методом проточной цитометрии определяли, подавляют ли EGFR-TfR -биспецифические антитела связывание трансферрина с TfR на раковых клетках.
Клетки OE21 высевали в лунки (по 1 х 106 клеток) 96-луночного планшета c U-образным дном (производство Falcon, Inc.) со средой RPMI 1640 (производство Sigma-Aldrich Co. LLC), содержащей 10% FBS, добавляли испытываемое антитело в концентрации 10 мкг/мл и держали планшет на ледяной бане в течение 1 часа. После этого прибавляли мышиный трансферрин, меченный Alexa Fluor 488 (Jackson ImmunoResearch, Inc.), и оставляли планшет на ледяной бане еще на 1 час. Затем промывали 2 раза раствором SB, суспендировали клетки в том же растворе и измеряли интенсивность флуоресценции в каждой лунке с помощью проточного цитометра, как в Примере 9.
На фиг. 11(A)-11(D) представлены результаты, полученные с цетуксимабом, TfR1071, E08-TfR1071, E12-TfR1071 и TfR435 соответственно. Антитело TfR435, нейтрализующее TfR и служившее положительным контролем, подавляло связывание трансферрина с TfR, но специфичное к TfR антитело TfR1071 и EGFR-TfR-биспецифические антитела E08-TfR1071 и E12-TfR1071 не подавляли связывание трансферрина с TfR так же, как и цетуксимаб, служивший отрицательным контролем.
Из этих данных следует, что EGFR-TfR-биспецифические антитела по данному изобретению не способны нейтрализовать TfR и не подавляют связывание трансферрина с TfR на клеточной поверхности.
Пример 14. Установление механизма проявления эффективности EGFR-TfR-биспецифических антител как лекарственного средства (уровень EGFR, уровень TfR и сигнальный путь с участием EGFR в клетках)
Чтобы выяснить механизм подавления роста раковых клеток биспецифическими антителами по данному изобретению, методом вестерн-блоттинга определяли влияние на сигнальный путь с участием EGFR и уровень белка TfR при добавлении EGFR-TfR-биспецифических антител к раковым клеткам.
Клетки OE21 высевали в лунки (по 1 × 106 клеток) плоскодонного 6-луночного планшета (производство Falcon, Inc.) со средой RPMI 1640 (производство Sigma-Aldrich Co. LLC), содержащей 10% FBS, добавляли к ним испытываемое антитело и культивировали клетки при температуре 37°C в атмосфере, содержащей 5,0% углекислого газа в течение 24 часов. После этого клетки помещали на ледяную баню, промывали 1 раз раствором PBS(-) и затем прибавляли буферный раствор RIPA (Thermo Fisher, Inc.), содержащий ингибиторную смесь Protease/Phosphatase Inhibitor Cocktail (CST Japan, Co., Ltd.); полученный лизат собирали скребком для клеток.
Собранный лизат центрифугировали со скоростью 15 000 об/мин при температуре 4°C в течение 5 минут, собирали супернатант и, взяв его часть, определяли содержание белка с помощью набора Pierce 660 nm Protein Assay Kit (Thermo Fisher, Inc.). К супернатанту прибавляли буферный раствор для образцов (для SDS-PAGE, сконцентрированный в 6 раз и содержащий восстанавливающий агент (; производство Nacalai Tesque, Inc.); полученный в результате раствор в количестве, содержавшем 10 мкг белка, наносили на готовые полиакриламидные гели (ATTO, Inc.) и проводили электрофорез в течение 25-80 минут в аппарате для PAGE (производство ATTO, Inc.).
В качестве стандарта при электрофорезе использовали окрашенный белковый маркер Prestained Protein Size Marker III (Wako Pure Chemical Corporation). Гели, фильтровальную бумагу (ATTO, Inc.) и мембрану (ATTO, Inc.) помещали в блоттер (ATTO, Inc.) и осуществляли блоттинг в течение 1 часа при токе 125 мА/гель. После блотирования мембрану держали в блокирующем буферном растворе (Pierce, Inc.) на шейкере при комнатной температуре в течение 1 часа или оставляли на ночь при температуре 4°C.
На мембрану наносили первичные антитела к различным белкам (биотинилированные антитела к Akt-киназе, биотинилированные антитела к фосфорилированной Akt-киназе, биотинилированные антитела к EGFR, биотинилированные антитела к фосфорилированному EGFR, кроличьи антитела к CD71; все – производства Cell Signaling Trechnology, Inc.) и держали на шейкере при комнатной температуре в течение 1 часа или оставляли на ночь при температуре 4°C.
Мембрану промывали 3 раз раствором TBS-T (Wako Pure Chemical Corporation), и наносили вторичные антитела (антитела, конъюгированные с пероксидазой хрена и стрептавидином, или антитела к кроличьему IgG, конъюгированные с пероксидазой хрена; Cell Signaling Trechnology, Inc.) к каждому из первых антител и держали на шейкере при комнатной температуре в течение 1 часа. Затем промывали 3 раза раствором TBS-T, добавляли реагент ECL Prime Western Blotting Detection Regent (GE Healthcare, Inc.), и сканировали мембрану на приборе Amersham Imager 600 (GE Healthcare, Inc.).
Как видно из фиг. 12, в случае антитела E08-TfR1071 количество фосфорилированного EGFR и фосфорилированной AKT-киназы снижалось так же, как в случае цетуксимаба, который служил положительным контролем, тогда как в случае E12-TfR1071 этого снижения не наблюдалось. А количество рецептора траснферрина снижалось в случае E08-TfR1071 и в случае E12-TfR1071, но не снижалось в случае цетуксимаба.
Эти результаты демонстрируют, что антитело E08-TfR1071 способно подавлять сигнальный путь с участием EGFR и вызывать распад TfR, а E12-TfR1071 не подавляет сигнальный путь с участием EGFR, обладая только способностью вызывать распад TfR .
Пример 15. Определение влияния EGFR-TfR-биспецифических антител
на экспрессию TfR
Чтобы выяснить, каким образом биспецифические антитела подавляют рост раковых клеток, определяли влияние EGFR-TfR -биспецифических антител на экспрессию TfR на клеточной мембране, как описано ниже.
Клетки OE21 высевали в лунки (по 1 × 106 клеток) плоскодонного 6-луночного планшета (производство Falcon, Inc.) со средой RPMI 1640 (производство Sigma-Aldrich Co. LLC), содержащей 10% FBS, добавляли испытываемое антитело в концентрации 10 мкг/мл и инкубировали при температуре 37°C в течение 24 часов. Клетки отделяли, как описано в Примере 9, прибавляли к ним трансферрин из сыворотки человеческой крови, конъюгированный с флуоресцентным красителем Alexa Fluor 488 (Molecular Probes, Inc.), в концентрации 5 мкг/мл и полученную смесь оставляли на 30 минут при температуре 4°C, после чего промывали, как описано в Примере 9. Количество трансферрина, связавшегося на поверхности клеток, рассчитывали по интенсивности флуоресценции одиночных клеток, измеренной с помощью проточного флуориметра.
На фиг. 13(A)-13(D) представлены результаты, полученные с антителами TfR1071, E08-TfR1071-биспецифическими антителами и E12-TfR1071-биспецифическими антителами соответственно. Как видно из фиг. 13, добавление EGFR-TfR-биспецифических антител вызывало уменьшения количества трансферрина, связанного на клеточной поверхности, тогда как в случае антитела TfR1071 этого не наблюдалось.
Эти результаты свидетельствуют, что подавление пролиферации раковых клеток EGFR-TfR-биспецифическими антителами по данному изобретению обусловлено распадом TfR, что отражается в уменьшении количества этого белка на поверхности указанных клеток.
Пример 16. Определение влияния EGFR-TfR-биспецифических антител
на поглощение клетками железа
Чтобы выяснить, какова роль железа в подавлении пролиферации раковых клеток EGFR-TfR-биспецифическими антителами, проделывали следующее.
Клетки OE21 высевали в лунки (по 1 × 103 клеток) плоскодонного 96-луночного планшета (производство Falcon, Inc.) со средой RPMI 1640 (производство Sigma-Aldrich Co. LLC), содержащей 10% FBS, добавляли двойную сернокислую соль трехвалентного железа и аммония (Nacalai Tesque, Inc., далее в настоящем документе обозначается FAS) до конечной концентрации 10 мкМ вместе с испытываемым антителом и культивировали клетки при температуре 37°C в атмосфере, содержащей 5,0% углекислого газа, в течение 4-6 суток. После этого определяли уровень ATP в жизнеспособных клетках, как описано в Примере 10.
Как видно из фиг. 14, если FAS не добавляли, то клеточный рост подавляли цетуксимаб, антитела TfR435, нейтрализующие TfR , и E12-TfR107-биспецифические антитела. Если же FAS добавляли, то антипролиферативная активность цетуксимаба не изменялась, тогда как антитела TfR435, нейтрализующие TfR , и E12-TfR107-биспецифические антитела ее не проявляли.
При добавлении к клеткам FAS железо поступает внутрь клеток без участия TfR. Поэтому в присутствии FAS не происходит истощения внутриклеточного запаса железа, сопровождающего блокирование TfR нейтрализующими его антителами или распад TfR под влиянием EGFR-TfR-биспецифических антител, так что подавления клеточного роста с участием TfR нет. Напротив, на подавлении клеточного роста, обусловленном ингибированием сигнального пути с участием эпидермального фактора роста, не сказывается поступление в клетки железа благодаря FAS. Таким образом, показано, что способность антител E12-TfR1071 подавлять клеточный рост обусловлена не ингибированием сигнального пути с участием EGFR, а отсутствием поступления железа в клетки – так же, как в случае антител, нейтрализующих TfR ..
Пример 17. Сравнение эффективности EGFR-TfR–биспецифических антител и ингибитора сигнального пути с участием EGFR
Чтобы сравнить два механизма подавления клеточного роста – ингибирование сигнального пути с участием EGFR и ингибирование TfR – были проведены эксперименты с использованием системы прижизненного анализа клеток IncuCyte (Sartorius AG).
Клетки OE21 высевали в лунки (по 1 × 103 клеток) плоскодонного 96-луночного планшета со средой RPMI 1640 (производство Sigma-Aldrich Co. LLC), содержащей 10% FBS, и вносили испытываемое антитело, разведенное той же средой, до концентрации 10 мкг/мл; клетки инкубировали при температуре 37°C в течение 5 суток; при этом раз в 2 часа делали фотографии с помощью системы IncuCyte (три поля зрения в каждой лунке). Заменяли среду, добавляли испытываемое антитело (концентрация 10 мкг/мл) и инкубировали еще 2 суток в тех же условиях. Еще раз заменяли среду и инкубировали клетки 7 суток в тех же условиях, но без испытываемого антитела. Для каждой точки фотографирования анализировали полученные данные с помощью программного обеспечения IncuCyte.
Как видно из фиг. 15, в случае цетуксимаба клеточный рост в среде без антитела возобновлялся, тогда как в случае биспецифического антитела E08-TfR1071 или E12-TfR1071 этого не наблюдалось. Когда к клеткам добавляли антитело TfR435 являющееся нейтрализующим по отношению к TfR, подавление клеточного роста сохранялось в свежей среде без антитела, хотя наблюдалась тендения к возобновлению клеточного роста;.; предполагается, что сильное подавление клеточного роста, сохраняющееся в отстутствие антител, в случае E08-TfR1071 или E12-TfR1071 обусловлено тем, что эти биспецифические антитела вызывают деградацию TfR .
Полученные результаты демонстрируют, что при ингибировании TfR подавление клеточного роста сохраняется даже после исчезновения ингибитора в отличие от подавления сигнального пути с участием EGFR.
Пример 18. Влияние EGFR-TfR-биспецифических антител
на человеческие клетки костного мозга.
Для определения влияния биспецифических антител E08-TfR1071 и E12- TfR1071 на здоровые клетки костного мозга человека (в них нет EGFR, но есть TfR ), проделывали следующее.
Человеческие клетки костного мозга (AllCells, Inc.) высевали в лунки (по 7500 клеток) 96-луночного планшета (производство Falcon, Inc.) со средой StemSpan (торговое название) SFEM II (STEMCELL Technologies, Inc.), добавляли испытываемое антитело и культивировали клетки при температуре 37°C в атмосфере, содержащей 5,0% углекислого газа, в течение 5 суток. После этого использовали набор для люминесцентного анализа жизнеспособности клеток CellTiter-Glo (зарегистрированное торговое название) Luminescent Cell Vialbility Assay (производство Promega Corporation) и измеряли интенсивность флуоресценции с помощью ридера для микропланшетов (1420 ARVO Multi-Label Counter, производство WALLAC, Inc.). Для каждого варианта условий отводили по 2 лунки и рассчитывали среднее значение. Полученная для данной лунки интенсивность флуоресценции отражает уровень ATP в жизнеспособных клетках в этой лунке
Чтобы вызвать дифференцировку человеческих клеток костного мозга в зрелые полипотентные гемопоэтические стволовые клетки (далее в настоящем документе обозначаются GEMM) в процессе культивирования к клеткам добавляли рекомбинантный человеческий фактор стволовых клеток (SCF) (PeproTech, Inc.) в конечной концентрации 50 нг/мл, человеческий интерлейкин-3 (IL-3) (Miltenyi Biotec, Inc.) в конечной концентрации 10 нг/мл, человеческий интерлейкин-6 (IL-6) (Sigma-Aldrich Co. LLC) в конечной концентрации 20 нг/мл, человеческий колониестимулирующий фактор гранулоцитов и макрофагов (GМ-CSF; Miltenyi Biotec, Inc.) в конечной концентрации 20 нг/мл, человеческий колониестимулирующий фактор гранулоцитов (G-CSF; Miltenyi Biotec, Inc.) в конечной концентрации 20 нг/мл, человеческий лиганд fms-подобной рецепторной тирозинкиназы 3 (Flt3-LG; Miltenyi Biotec, Inc.) в конечной концентрации 50 нг/мл, эритропоэтин (Kyowa Hakko Kirin Co., Ltd.) в конечной концентрации 3 Ед/мл и человеческий тромбопоэтин (TPO; Miltenyi Biotec, Inc.) в конечной концентрации 30 нг/м.
Как видно из фиг. 16, антитела TfR435, нейтрализующие TfR , подавляют клеточный рост, а биспецифические антитела E08-TfR1071 и E12-TfR1071 не проявляют такой активности.
Эти результаты подтверждают, что биспецифические антитела по данному изобретению не способны подавлять пролиферацию здоровых клеток костного мозга, в которых экспрессируется TfR . Следовательно, биспецифические антитела по данному изобретению должны быть менее токсичны для костного мозга, чем антитела, нейтрализующие TfR .
19. Способность подавлять клеточный рост у EGFR-TfR-биспецифических антител с изменениями в аминокислотной последовательности
Способность биспецифических антител E12-TfR1071, у которых внесены изменения в аминокислотную последовательность VH TfR1071, подавлять клеточный рост определяли, как описано в Примере 12, используя клетки OE21. Вначале получили EGFR-TfR-биспецифические антитела с изменениями в аминокислотной последовательности; для этого проделывали следующее. Чтобы получить экспрессионный вектор для биспецифических антител с изменениями в аминокислотной последовательности (с аминокислотными заменами), в экспрессионном векторе, в котором содержались нуклеотидные последовательности, кодирующие аминокислотные последовательности антитела E12-TfR1071 без изменений, заменили их на нуклеотидные последовательности, кодирующие аминокислотные последовательности с заменами, как описано в Примере 6. Экспрессия и получение биспецифических антител с аминокислотными заменами осуществляли, как описано в Примере 7. Были получены следующие биспецифические антитела с заменами аминокислотных остатков.
Модифицированные антитела TfR1071: Y32A, Y32F, T33A, T33G, L45A, V48A, V50A, I51A, I51L, N52A-A, N52A-D, V55E, D58A, D61P, Q97A, Q97D, P98A, W99A, W99F, W99H, W99Y, Y100A-A, Y100A-F, V102L, P98Y, P98S, P98D, P98Q, P98E, P98T, P98R, P98G, P98K, P98M, P98V, P98L, P98I, P98W, P98F, P98H
В каждом из приведенных выше обозначений указана замена аминокислотного остатка в модифицированном антителе, а именно: [однобуквенное обозначение аминокислоты до замены] [положение данного аминокислотного остатка в аминокислотной последовательности VH (SEQ ID NO: 31) антитела TfR1071 по индексу EU] [однобуквенное обозначение аминокислоты после замены]. Далее в настоящем документе аминокислотные замены обозначаются таким же образом.
Например, обозначение Y32A означает, что остаток тирозина (обозначается буквой Y) в положении 32 (согласно индексу EU) заменен на остаток аланина (обозначается буквой А). Обозначения 52A и 100A означают, что данный аминокислотный остаток находится в положении «52-53» и «100-101» соответственно (по индексу EU). Например, обозначение N52A-D означает, что в положении 52A остаток согласно индексу EU заменен с N на А, а обозначение Y100A-A означает, что в положении 100A согласно индексу EU изменен с Y на А.
Раковые клетки высевали в лунки (по 1 × 103 клеток) плоскодонного 96-луночного планшета (производство Falcon, Inc.), добавляли испытываемое антитело, разведенное до различных концентраций средой RPMI 1640 (производство Sigma-Aldrich Co. LLC), содержащей 10% FBS, и культивировали клетки при температуре 37°C в атмосфере, содержащей 5,0% углекислого газа в течение 4-6 суток. После этого прибавляли реагент набора Cell Counting Kit-8 (Dojindo, Inc.) и измеряли поглощение с помощью микропланшетного ридера (1420 ARVO Multi-Label Counter, производство WALLAC, Inc.). Для каждого варианта условий отводили по 3 лунки и рассчитывали среднее значение. Полученная для данной лунки интенсивность флуоресценции отражает количество жизнеспособных клетках в этой лунке. В качестве отрицательного контроля использовали антитела к DNP. Результаты влияния указанных выше биспецифических антител на клеточный рост представлены на фиг. 17A и 17B.
Как видно из фиг. 17A и 17B, модифицированные антитела клона TfR1071 с аминокислотными заменами N52A-A и N52A-D не проявляли способность подавлять клеточный рост, модифицированные антитела с аминокислотными заменами V55E, W99A и W99H обладали такой способностью в малой степени, а у всех остальных модифицированных антител наблюдалась высокая способность подавлять клеточный рост в сравнении с таковой до изменения аминокислотной последовательности.
Эти результаты демонстрируют, что в биспецифических антителах E12-TfR1071 по данному изобретению N52 важен для способности подавлять клеточный рост. Далее даже если остаток P98 заменен на остаток любой другой аминокислоты наблюдается эквивалентная способность антитела подавлять клеточный рост; предположительно, этот аминокислотный остаток может быть заменен остатком любой другой природной аминокислоты.
Пример 20. Идентификация эпитопа,
распознаваемого антителом к TfR
Эпитоп, распознаваемый антителом к TfR 1071, идентифицировали следующим образом.
Способом, описанным в Примере 7, на основании сведений о последовательностях, приведенных в публикации WO 2014/189973, были получены антитела к TfR 15G11v5, 7A4v15 и 16F6v4, которые связываются с апикальным доменом человеческого TfR, и 7G7v1, которое распознает его протеазоподобный домен
Был получен внеклеточный домен человеческого TfR, описанный в Примере 1. Далее получили химерный белок, в котором апикальный либо протеазоподобный домен внеклеточного домена человеческого TfR был заменен на соответствующий домен мышиного TfR (содержащий аминокислотную последовательность, представленную в базе данных GenBank под регистрационным номером NP_001344227, или аминокислотную последовательность, представленную SEQ ID NO: 87). А именно, нуклеотидную последовательность., кодирующую эту аминокислотную последовательность, субклонировали в векторе pCI-OtCM, в который включили гистидиновый тэг; таким образом получился экспрессионный вектор для желаемого белка. Используя этот вектор, способом. описанным в Примере 1, получили белки His-huTfR, huTfR с мышиным апикальным доменом (SEQ ID NO: 88) и huTfR с мышиным протеазоподобным доменом (SEQ ID NO: 89), в каждый из которых был добавлен гистидиновый тэг.
Обозначение His-huTfR представляет внеклеточный домен человеческого TfR с гистидиновым тэгом; название «huTfR с мышиным апикальным доменом» представляет внеклеточный домен человеческого TfR с гистидиновым тэгом, в котором апикальный домен заменен на соответствующую мышиную последовательность; название «huTfR с мышиным протеазоподобным доменом» представляет внеклеточный домен человеческого TfR с гистидиновым тэгом, в котором протеазоподобный домен заменен на соответствующую мышиную последовательность.
Аналогично были получены химерные TfR A01-A16, в которых некоторые аминокислотные остатки в апикальном домене человеческого TfR заменены соответственными аминокислотными остатками, свойственными мышиному TfR. Полученные химерные белки представлены в таблице 5,; их аминокислотные последовательности представлены SEQ ID NO: 90-105 соответственно.
В таблице 5 для каждого химерного белка указаны сделанные аминокислотные замены. Например, в случае белка А01 запись в правом столбце таблицы Q285K-T286N-T362S означает, что в аминокислотной последовательности человеческого TfR, представленной SEQ ID NO: 6, в положении 285 аминокислотный остаток Q заменен на K, в положении 286 аминокислотный остаток T) заменен на N и в положении 362 аминокислотный остаток T заменен на S. В тех случаях, когда нет соответствия остатка в мышином TfR, V210 of A08, например V210 или A08, то аминокислоту удаляли.
Таблица 5. Химерные белки TfR для идентификации эпитопа
Затем провели анализ эпитопов, для различных антителам к TfR. Сначала методом проточной цитометрии, как описано в Примере 9, определяли реакционноспособность антител к TfR, продуцируемых человеческими и мышиными клетками TfR/CHO, полученными, как описано в Примере 2. Оказалось, что антитела R327, TfR1071, cyno186, cyno292 и 2230 связываются с человеческим TfR, но не связываются с мышиным. Кроме того, из публикации WO 2014/189973 известно, что у антител 15G11v5, 7A4v15, 16F6v4 и 7G7v1 отсутствует перекрестная реактивность по отношению к мышиному белку.
Затем методом ELISA определяли активность связывания антител к TfR с различными химерными белками TfR, полученными, как описано выше; таким образом были идентифицированы связывающий домен антител к TfR и распознаваемый ими эпитоп. Если с химерным белком TfR связывания с антителами не наблюдалось, считалось, что тот аминокислотный остаток, который в данном химерном белке заменен, входит в состав эпитопа.
ELISA проводили следующим образом. На 96-луночном иммунологическом планшете (NUNC, Inc.) иммобилизовали антигенный белок, блокировали неспецифическое связывание 1%-ным (масса/объем) раствором BSA-PBS(-) с pH 7,0, не содержащим KCl (далее в настоящем документе называется блокирующим раствором; Nacalai Tesque, Inc.) при комнатной температуре, после чего вносили антитело к TfR, разведенное тем же раствором и для протекания реакции оставляли на 1 час при комнатной температуре. Затем промывали 0,05%-ным (масса/объем) раствором -tPBS (1x) без KCl (pH 7,2) (далее в настоящем документе называется раствором для промывания; Nacalai Tesque, Inc.), добавляли анти- IgG человека (Fc) козьи IgG Fab”-HRP (IBL, Inc),. разведенные в блокирующем растворе, и оставляли на 1 час при комнатной температуре для протекания реакции. Промывали раствором для промывания, прибавляли раствор субстрата 1-Step (зарегистрированное торговое название) Ultra TMB-ELISA Substrate Solution (Thermo, Inc.) и оставляли при комнатной температуре для проявления окрашивания. Цветную реакцию останавливали добавлением раствора соляной кислоты (5 н) и измеряли поглощение на длине волны 450 нм (референсная длина волны 570 нм) с помощью планшетного ридера (EPOCH 2; BioTek Instruments, Inc.).
Полученные результаты представлены в таблице 6 (обозначение N/A означает, что данных нет). Как меру активности связывания с антигенным белком брали разницу в значениях поглощения на длине волны 450 нм и на референсной длине волны 570 нм; те случаи, когда величина, полученная от деления значения активности связывания с тем или иным химерным TfR на значение активности связывания с His-huTfR, была 0,5 или больше, обозначены (++); те случаи, когда указанная величина была 0,2 или больше, но меньше 0,5, обозначены (+); те случаи, когда эта величина была меньше 0,2, обозначены (-).
Таблица 6. Определение эпитопов для различных антител к TfR
к TfR
антигенным белком
по данным ELISA
химерным белком TfR
по данным ELISA
в апикальном
домене
с мышиным
апикальным доменом
с мышиным
протеазо-
подобным доменом
Эти результаты демонстрируют, что антитела T14, R327, TfR1071, cyno186, cyno292, 15G11v5, 7A4v15 и 16F6v4 распознают апикальный домен TfR, а антитела 2230 и 7G7v1 - протеазоподобный домен. Из числа антител, распознающих апикальный домен, антитело T14 распознает сайты аминокислотных замен в A05; R327, TfR1007, TfR1071, cyno292 - A02 и A07; cyno186 - в A02, A07 и A14; 15G11v5 – в A02, A03 и A04; 7A4v15 и 16F6v4 – в A02.
Показано, что аминокислотные остатки, распознаваемые антителом TfR1071, - это те, которые заменены в химерных белках A02 и A07. Таким образом, можно полагать, что антитело TfR1071 распознает семь аминокислотных остатков, а именно D352, S355, D356, K358, M365, V366 и E369. То же можно сказать об антителах R327, TfR1007 и cyno292.
Пример 21. Эпитоп, распознаваемый антителом к TfR, и способность EGFR-TfR-биспецифических антител подавлять клеточный рост
Чтобы изучить взаимосвязь между способностью биспецифических антител по данному изобретению подавлять клеточный рост и паратопом вариабельной области части IgG, который связывается с эпитопом TfR, были получены биспецифические антитела E12-TfR, для которых использовались различные антитела к TfR, распознающие различные эпитопы, и определена их способность подавлять клеточный рост.
Легкая цепь антител к TfR 15G11v5, 7A4v15, 16F6v4 и 7G7v1 и легкая цепь антитела к EGFR E12 различаются, поэтому были получены биспецифические антитела, имеющие структуру, изображенную на фиг. 18(A), для чего применили способ, описанный в Примере 6. Для определения активности связывания биспецифических антител действовали, как описано в Примере 8, а именно на сенсорном чипе СМ5 иммобилизовали антитела для выявления белков с гистидиновым тэгом Penta-His Antibody (QIAGEN, Inc.), прибавляли белок His-huTfR и определяли активность связывания аналита – биспецифического антитела; для каждого из биспецифических антител рассчитывали константы ассоциации/диссоциации и константу равновесия. Результаты представлены в таблице 7.
Таблица 7. Активность связывания биспецифических антител с TfR
Как видно из таблицы 7, все полученные биспецифические антитела прочно связывались с TfR .
Результаты определения подавления пролиферации клеток ОЕ21 полученными биспецифическими антителами, которое проводили, как описано в Примере 19, изображены на фиг. 19A и 19B.
Как видно из фиг. 19A и 19B, максимальная активность у EGFR-TfR-биспецифических антител на основе антител R327, TfR1007, TfR1071, cyno186 и cyno292 была выше, чем у остальных биспецифических антител. Судя по этим результатам и по результатам Примера 20, высокой способностью подавлять клеточный рост обладают биспецифические антитела на основе антител R327, TfR1007, TfR1071 и cyno292, распознающих эпитопы, в структуре которых участвуют сайты аминокислотных замен в белках A02 и A07, а также cyno186, распознающего эпитопы, в структуре которых участвуют сайты аминокислотных замен в белках A02, A07 и A14. Таким образом. показано, что для высокой способности подавлять клеточный рост нужно, чтобы антитело распознавало эпитоп, образуемый с участием аминокислотных остатков, замененных в белках A02 и A07.
Кроме того, как видно из фиг. 19A, среди тех биспецифических антител, которые распознают эпитоп, образуемый с участием аминокислотных остатков. замененных в белках A02 и A07, наибольшей активностью начиная с наименьшей концентрации обладали биспецифические антитела на основе антител TfR1071 и R327. Иными словами, клоны TfR1071 и R327 оказались наилучшими.
Пример 22. Подавление клеточного роста EGFR-TfR-биспецифическими антителами на основе различных антител к EGFR и антитела TfR1071
Чтобы выяснить, действительно ли компонент TfR1071 важен для способности биспецифических антител подавлять клеточный рост, были получены EGFR-TfR-биспецифические антитела, в которых использовались имеющиеся в продаже антитела к EGFR, способные ингибировать этот рецептор, и антитело к TfR TfR1071, и определена их способность подавлять клеточный рост.
Были получены биспецифические антитела из антител к EGFR (цетуксимаба, панитумумаба, нецитумумаба и нимотузумаба) и антитела к TfR TfR1071, как описано в Примере 6. Использовали последовательности вариабельных областей цетуксимаба, панитумумаба, нецитумумаба и нимотузумаба, описанные в публикациях WO 1996/040210, WO 1998/050433, WO 2011/116387 и в заявке на патент США № 2012/0308576 соответственно. Структура этих антител соответствует изображенной на фиг. 1(C).
Результаты определения подавления клеточного роста полученными биспецифическими антителами, показателем чего служило подавление пролиферации раковых клеток по Примеру 20, изображены на фиг. 20.
Как видно из фиг. 20, высокую способность подавлять клеточный рост проявляли EGFR-TfR-биспецифические антитела, в которых имелись CDR антитела TfR1071, а вариабельная область могла быть из любого антитела к EGFR.
На основании этих результатов можно полагать, что для того, чтобы биспецифическое антитело обладало высокой способностью подавлять клеточный рост, в его части IgG должны содержаться CDR антитела TfR1071.
Пример 23. Сравнение способности гетеродимерных биспецифических антител подавлять клеточный рост
Чтобы выяснить, какое значение имеет валентность связывания биспецифических антител по данному изобретению с TfR и с EGFR для их способности подавлять клеточный рост, было получено гетеродимерное биспецифическое антитело, в котором имелись один антиген-связывающий домен антитела E12 к EGFR и один антиген-связывающий домен антитела TfR1071 к TfR (далее в настоящем документе оно называется гетеродимерным антителом), и определена его способность подавлять клеточный рост. На фиг. 18(B) схематически изображена структура этого гетеродимерного антитела.
Гетеродимерное биспецифическое антитело Hetero E12-TfR107, в котором имелись один антиген-связывающий домен антитела E12 к EGFR и один антиген-связывающий домен антитела TfR107 к TfR, получали способом, описанным в патентной литературе (см. заявку на патент США № 2014/0348839); его способность подавлять клеточный рост определяли, как описано в Примере 11. Результаты представлены на фиг. 21.
Как видно из фиг. 21, максимальная активность и удельная активность подавления клеточного роста у Hetero E12-TfR1071 слабее, чем у биспецифического антитела E12-TfR1071. Полученные результаты демонстрируют, что биспецифические антитела по данному изобретению, у которых валентность по отношению к каждому из антигенов – к TfR и к EGFR - равна 2, обладают более высокой способностью подавлять клеточный рост, нежели гетеродимерные антитела, у которых валентность по отношению к каждому из антигенов – к TfR и к EGFR – равна 1.
Пример 24. Активность связывания
биспецифического антитела E12-TfR1071YE с TfR
Биспецифическое антитело E12-TfR1071YE, имеющее VH последовательность (SEQ ID NO: 106), в которой аминокислота в положении 1 VH последовательности (SEQ ID NO: 31) TfR1071 заменена с тирозина (Y) на (E (глутаминовую кислоту.Определяли активность связывания этого биспецифического антитела с человеческим TfR методом ELISA, как описано в Примере 20. Активность связывания биспецифического антитела E12-TfR1071YE оказалась сравнимой с таковой E12-TfR1071.
Данное изобретение описано в настоящем документе подробно в его конкретных аспектах, однако специалисту в данной области техники очевидно, что возможны различные изменения и модификации, не выходящие за пределы сущности и объема данного изобретения. Настоящая заявка основывается на заявке на патент Японии-, поданной 28 декабря 2018 г. (заявка на патент № 2018-248334), которая полностью включается в настоящий документ путем отсылки.
Перечень последовательностей
SEQ ID NO: 1: нуклеотидная последовательность внеклеточного домена человеческого TfR
SEQ ID NO: 2: аминокислотная последовательность внеклеточного домена человеческого TfR
SEQ ID NO: 3: нуклеотидная последовательность внеклеточного домена обезьяньего TfR
SEQ ID NO: 4: аминокислотная последовательность внеклеточного домена обезьяньего TfR
SEQ ID NO: 5: нуклеотидная последовательность человеческого TfR
SEQ ID NO: 6: аминокислотная последовательность человеческого TfR
SEQ ID NO: 7: нуклеотидная последовательность TfR яванского макака
SEQ ID NO: 8: аминокислотная последовательность TfR яванского макака
SEQ ID NO: 9: нуклеотидная последовательность внеклеточного домена человеческого EGFR (включая сигнальную последовательность)
SEQ ID NO: 10: аминокислотная последовательность внеклеточного домена человеческого EGFR (включая сигнальную последовательность)
SEQ ID NO: 11: нуклеотидная последовательность внеклеточного домена обезьяньего EGFR (включая сигнальную последовательность)
SEQ ID NO: 12: аминокислотная последовательность внеклеточного домена обезьяньего EGFR (включая сигнальную последовательность)
SEQ ID NO: 13: нуклеотидная последовательность человеческого EGFR
SEQ ID NO: 14: аминокислотная последовательность человеческого EGFR
SEQ ID NO: 15: нуклеотидная последовательность VL A27
SEQ ID NO: 16: аминокислотная последовательность VL A27
SEQ ID NO: 17: аминокислотная последовательность LCDR1 A27
SEQ ID NO: 18: аминокислотная последовательность LCDR2 A27
SEQ ID NO: 19: аминокислотная последовательность LCDR3 A27
SEQ ID NO: 20: нуклеотидная последовательность VH PSM4072
SEQ ID NO: 21: аминокислотная последовательность VH PSM4072
SEQ ID NO: 22: аминокислотная последовательность HCDR1 PSM4072
SEQ ID NO: 23: аминокислотная последовательность HCDR2 PSM4072
SEQ ID NO: 24: аминокислотная последовательность HCDR3 PSM4072
SEQ ID NO: 25: нуклеотидная последовательность VH R327
SEQ ID NO: 26: аминокислотная последовательность VHR327
SEQ ID NO: 27: аминокислотная HCDR1 R327
SEQ ID NO: 28: аминокислотная последовательность HCDR2 R327
SEQ ID NO: 29: аминокислотная последовательность HCDR3 R327
SEQ ID NO: 30: нуклеотидная последовательность VH TfR1071
SEQ ID NO: 31: аминокислотная последовательность VH TfR1071
SEQ ID NO: 32: аминокислотная последовательность HCDR1 TfR1071
SEQ ID NO: 33: аминокислотная последовательность HCDR2 TfR1071
SEQ ID NO: 34: аминокислотная последовательность HCDR3 TfR1071
SEQ ID NO: 35: нуклеотидная последовательность VH TfR4016
SEQ ID NO: 36: аминокислотная последовательность VH TfR4016
SEQ ID NO: 37: аминокислотная последовательность HCDR1 TfR4016
SEQ ID NO: 38: аминокислотная последовательность HCDR2 TfR4016
SEQ ID NO: 39: аминокислотная последовательность HCDR3 TfR4016
SEQ ID NO: 40: нуклеотидная последовательность VH cyno186
SEQ ID NO: 41: аминокислотная последовательность VH cyno186
SEQ ID NO: 42: аминокислотная последовательность HCDR1 cyno186
SEQ ID NO: 43: аминокислотная последовательность HCDR2 cyno186
SEQ ID NO: 44: аминокислотная последовательность HCDR3 cyno186
SEQ ID NO: 45: нуклеотидная последовательность VH cyno292
SEQ ID NO: 46: аминокислотная последовательность VH cyno292
SEQ ID NO: 47: аминокислотная последовательность HCDR1 cyno292
SEQ ID NO: 48: аминокислотная последовательность HCDR2 cyno292
SEQ ID NO: 49: аминокислотная последовательность HCDR3 cyno292
SEQ ID NO: 50: аминокислотная последовательность VH TfR1007
SEQ ID NO: 51: аминокислотная последовательность VH cyno163
SEQ ID NO: 52: аминокислотная последовательность VH 2230
SEQ ID NO: 53: аминокислотная последовательность VL2230
SEQ ID NO: 54: аминокислотная последовательность VH T14
SEQ ID NO: 55: аминокислотная последовательность VH TfR434
SEQ ID NO: 56: аминокислотная последовательность VH TfR435
SEQ ID NO: 57: аминокислотная последовательность VL TfR434 и TfR435
SEQ ID NO: 58: нуклеотидная последовательность VH E08
SEQ ID NO: 59: аминокислотная последовательность VH E08
SEQ ID NO: 60: аминокислотная последовательность HCDR1 E08
SEQ ID NO: 61: аминокислотная последовательность HCDR2 E08
SEQ ID NO: 62: аминокислотная последовательность HCDR3 E08
SEQ ID NO: 63: нуклеотидная последовательность VH E12
SEQ ID NO: 64: аминокислотная последовательность VH E12
SEQ ID NO: 65: аминокислотная последовательность HCDR1 E12
SEQ ID NO: 66: аминокислотная последовательность HCDR2 E12
SEQ ID NO: 67: аминокислотная последовательность HCDR3 E12
SEQ ID NO: 68: нуклеотидная последовательность VH E17
SEQ ID NO: 69: аминокислотная последовательность VH E17
SEQ ID NO: 70: аминокислотная последовательность HCDR1 E17
SEQ ID NO: 71: аминокислотная последовательность HCDR2 E17
SEQ ID NO: 72: аминокислотная последовательность HCDR3 E17
SEQ ID NO: 73: аминокислотная последовательность VH KME07
SEQ ID NO: 74: аминокислотная последовательность VH KME09
SEQ ID NO: 75: аминокислотная последовательность VH KME11
SEQ ID NO: 76: аминокислотная последовательность VH цетуксимаба
SEQ ID NO: 77: аминокислотная последовательность VL цетуксимаба
SEQ ID NO: 78: аминокислотная последовательность VH HN3
SEQ ID NO: 79: аминокислотная последовательность линкера
SEQ ID NO: 80: аминокислотная последовательность линкера
SEQ ID NO: 81: аминокислотная последовательность линкера
SEQ ID NO: 82: аминокислотная последовательность линкера
SEQ ID NO: 83: нуклеотидная последовательность константной области IgG4PE R409K
SEQ ID NO: 84: аминокислотная последовательность константной области IgG4PE R409K
SEQ ID NO: 85: нуклеотидная последовательность с модифицированными кодонами области СН1 IgG4
SEQ ID NO: 86: аминокислотная последовательность константной области IgG4PE
SEQ ID NO: 87: аминокислотная последовательность мышиного TfR
SEQ ID NO: 88: аминокислотная последовательность химерного белка, включающего внеклеточный домен человеческого TfR и апикальный домен мышиного TfR
SEQ ID NO: 89: аминокислотная последовательность химерного белка, включающего внеклеточный домен человеческого TfR и протеазоподобный домен мышиного TfR
SEQ ID NO: 90: аминокислотная последовательность A01
SEQ ID NO: 91: аминокислотная последовательность A02
SEQ ID NO: 92: аминокислотная последовательность A03
SEQ ID NO: 93: аминокислотная последовательность A04
SEQ ID NO: 94: аминокислотная последовательность A05
SEQ ID NO: 95: аминокислотная последовательность A06
SEQ ID NO: 96: аминокислотная последовательность A07
SEQ ID NO: 97: аминокислотная последовательность A08
SEQ ID NO: 98: аминокислотная последовательность A09
SEQ ID NO: 99: аминокислотная последовательность A10
SEQ ID NO: 100: аминокислотная последовательность A11
SEQ ID NO: 101: аминокислотная последовательность A12
SEQ ID NO: 102: аминокислотная последовательность A13
SEQ ID NO: 103: аминокислотная последовательность A14
SEQ ID NO: 104: аминокислотная последовательность A15
SEQ ID NO: 105: аминокислотная последовательность A16
SEQ ID NO: 106: аминокислотная последовательность VH TfR1071 начиная с EVQL
SEQ ID NO: 107: аминокислотная последовательность HCDR1 TfR1007
SEQ ID NO: 108: аминокислотная последовательность HCDR2 TfR1007
SEQ ID NO: 109: аминокислотная последовательностьHCDR3 TfR1007
SEQ ID NO: 110: аминокислотная последовательность VH цетуксимаба
SEQ ID NO: 111: аминокислотная последовательность VL цетуксимаба
SEQ ID NO: 112: аминокислотная последовательность VH панитумумаба
SEQ ID NO: 113: аминокислотная последовательность VL панитумумабаа
SEQ ID NO: 114: аминокислотная последовательность VH нецитумумаба
SEQ ID NO: 115: аминокислотная последовательность VL нецитумумаба
SEQ ID NO: 116: аминокислотная последовательность VH нимотузумаба
SEQ ID NO: 117: аминокислотная последовательность VL нимотузумаба
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> Kyowa Kirin Co., Ltd.
<120> Bispecific antibody binding to TfR
<130> W528470
<150> JP2018-248334
<151> 2018-12-28
<160> 117
<170> PatentIn version 3.5
<210> 1
<211> 1643
<212> DNA
<213> Homo sapiens
<400> 1
tggagaatcc tgggggttat gtggcgtata gtaaggctgc aacagttact ggtaaactgg
60
tccatgctaa ttttggtact aaaaaagatt ttgaggattt atacactcct gtgaatggat
120
ctatagtgat tgtcagagca gggaaaatca cctttgcaga aaaggttgca aatgctgaaa
180
gcttaaatgc aattggtgtg ttgatataca tggaccagac taaatttccc attgttaacg
240
cagaactttc attctttgga catgctcatc tggggacagg tgacccttac acacctggat
300
tcccttcctt caatcacact cagtttccac catctcggtc atcaggattg cctaatatac
360
ctgtccagac aatctccaga gctgctgcag aaaagctgtt tgggaatatg gaaggagact
420
gtccctctga ctggaaaaca gactctacat gtaggatggt aacctcagaa agcaagaatg
480
tgaagctcac tgtgagcaat gtgctgaaag agataaaaat tcttaacatc tttggagtta
540
ttaaaggctt tgtagaacca gatcactatg ttgtagttgg ggcccagaga gatgcatggg
600
gccctggagc tgcaaaatcc ggtgtaggca cagctctcct attgaaactt gcccagatgt
660
tctcagatat ggtcttaaaa gatgggtttc agcccagcag aagcattatc tttgccagtt
720
ggagtgctgg agactttgga tcggttggtg ccactgaatg gctagaggga tacctttcgt
780
ccctgcattt aaaggctttc acttatatta atctggataa agcggttctt ggaaccagca
840
acttcaaggt ttctgccagc ccactgttgt atacgcttat tgagaaaaca atgcaaaatg
900
tgaagcatcc ggttactggg caatttctat atcaggacag caactgggcc agcaaagttg
960
agaaactcac tttagacaat gctgctttcc ctttccttgc atattctgga atcccagcag
1020
tttctttctg tttttgcgag gacacagatt atccttattt gggaaccacc atggacacct
1080
ataaggaact gattgagagg attcctgagt tgaacaaagt ggcacgagca gctgcagagg
1140
tcgctggtca gttcgtgatt aaactaaccc atgatgttga attgaacctg gactatgaga
1200
ggtacaacag ccaactgctt tcatttgtga gggatctgaa ccaatacaga gcagacataa
1260
aggaaatggg cctgagttta cagtggctgt attctgctcg tggagacttc ttccgtgcta
1320
cttccagact aacaacagat ttcgggaatg ctgagaaaac agacagattt gtcatgaaga
1380
aactcaatga tcgtgtcatg agagtggagt atcacttcct ctctccctac gtatctccaa
1440
aagagtctcc tttccgacat gtcttctggg gctccggctc tcacacgctg ccagctttac
1500
tggagaactt gaaactgcgt aaacaaaata acggtgcttt taatgaaacg ctgttcagaa
1560
accagttggc tctagctact tggactattc agggagctgc aaatgccctc tctggtgacg
1620
tttgggacat tgacaatgag ttt
1643
<210> 2
<211> 672
<212> PRT
<213> Homo sapiens
<400> 2
Cys Lys Gly Val Glu Pro Lys Thr Glu Cys Glu Arg Leu Ala Gly Thr
1 5 10 15
Glu Ser Pro Val Arg Glu Glu Pro Gly Glu Asp Phe Pro Ala Ala Arg
20 25 30
Arg Leu Tyr Trp Asp Asp Leu Lys Arg Lys Leu Ser Glu Lys Leu Asp
35 40 45
Ser Thr Asp Phe Thr Gly Thr Ile Lys Leu Leu Asn Glu Asn Ser Tyr
50 55 60
Val Pro Arg Glu Ala Gly Ser Gln Lys Asp Glu Asn Leu Ala Leu Tyr
65 70 75 80
Val Glu Asn Gln Phe Arg Glu Phe Lys Leu Ser Lys Val Trp Arg Asp
85 90 95
Gln His Phe Val Lys Ile Gln Val Lys Asp Ser Ala Gln Asn Ser Val
100 105 110
Ile Ile Val Asp Lys Asn Gly Arg Leu Val Tyr Leu Val Glu Asn Pro
115 120 125
Gly Gly Tyr Val Ala Tyr Ser Lys Ala Ala Thr Val Thr Gly Lys Leu
130 135 140
Val His Ala Asn Phe Gly Thr Lys Lys Asp Phe Glu Asp Leu Tyr Thr
145 150 155 160
Pro Val Asn Gly Ser Ile Val Ile Val Arg Ala Gly Lys Ile Thr Phe
165 170 175
Ala Glu Lys Val Ala Asn Ala Glu Ser Leu Asn Ala Ile Gly Val Leu
180 185 190
Ile Tyr Met Asp Gln Thr Lys Phe Pro Ile Val Asn Ala Glu Leu Ser
195 200 205
Phe Phe Gly His Ala His Leu Gly Thr Gly Asp Pro Tyr Thr Pro Gly
210 215 220
Phe Pro Ser Phe Asn His Thr Gln Phe Pro Pro Ser Arg Ser Ser Gly
225 230 235 240
Leu Pro Asn Ile Pro Val Gln Thr Ile Ser Arg Ala Ala Ala Glu Lys
245 250 255
Leu Phe Gly Asn Met Glu Gly Asp Cys Pro Ser Asp Trp Lys Thr Asp
260 265 270
Ser Thr Cys Arg Met Val Thr Ser Glu Ser Lys Asn Val Lys Leu Thr
275 280 285
Val Ser Asn Val Leu Lys Glu Ile Lys Ile Leu Asn Ile Phe Gly Val
290 295 300
Ile Lys Gly Phe Val Glu Pro Asp His Tyr Val Val Val Gly Ala Gln
305 310 315 320
Arg Asp Ala Trp Gly Pro Gly Ala Ala Lys Ser Gly Val Gly Thr Ala
325 330 335
Leu Leu Leu Lys Leu Ala Gln Met Phe Ser Asp Met Val Leu Lys Asp
340 345 350
Gly Phe Gln Pro Ser Arg Ser Ile Ile Phe Ala Ser Trp Ser Ala Gly
355 360 365
Asp Phe Gly Ser Val Gly Ala Thr Glu Trp Leu Glu Gly Tyr Leu Ser
370 375 380
Ser Leu His Leu Lys Ala Phe Thr Tyr Ile Asn Leu Asp Lys Ala Val
385 390 395 400
Leu Gly Thr Ser Asn Phe Lys Val Ser Ala Ser Pro Leu Leu Tyr Thr
405 410 415
Leu Ile Glu Lys Thr Met Gln Asn Val Lys His Pro Val Thr Gly Gln
420 425 430
Phe Leu Tyr Gln Asp Ser Asn Trp Ala Ser Lys Val Glu Lys Leu Thr
435 440 445
Leu Asp Asn Ala Ala Phe Pro Phe Leu Ala Tyr Ser Gly Ile Pro Ala
450 455 460
Val Ser Phe Cys Phe Cys Glu Asp Thr Asp Tyr Pro Tyr Leu Gly Thr
465 470 475 480
Thr Met Asp Thr Tyr Lys Glu Leu Ile Glu Arg Ile Pro Glu Leu Asn
485 490 495
Lys Val Ala Arg Ala Ala Ala Glu Val Ala Gly Gln Phe Val Ile Lys
500 505 510
Leu Thr His Asp Val Glu Leu Asn Leu Asp Tyr Glu Arg Tyr Asn Ser
515 520 525
Gln Leu Leu Ser Phe Val Arg Asp Leu Asn Gln Tyr Arg Ala Asp Ile
530 535 540
Lys Glu Met Gly Leu Ser Leu Gln Trp Leu Tyr Ser Ala Arg Gly Asp
545 550 555 560
Phe Phe Arg Ala Thr Ser Arg Leu Thr Thr Asp Phe Gly Asn Ala Glu
565 570 575
Lys Thr Asp Arg Phe Val Met Lys Lys Leu Asn Asp Arg Val Met Arg
580 585 590
Val Glu Tyr His Phe Leu Ser Pro Tyr Val Ser Pro Lys Glu Ser Pro
595 600 605
Phe Arg His Val Phe Trp Gly Ser Gly Ser His Thr Leu Pro Ala Leu
610 615 620
Leu Glu Asn Leu Lys Leu Arg Lys Gln Asn Asn Gly Ala Phe Asn Glu
625 630 635 640
Thr Leu Phe Arg Asn Gln Leu Ala Leu Ala Thr Trp Thr Ile Gln Gly
645 650 655
Ala Ala Asn Ala Leu Ser Gly Asp Val Trp Asp Ile Asp Asn Glu Phe
660 665 670
<210> 3
<211> 2016
<212> DNA
<213> Monkey
<400> 3
tgtaaagggg tagaaccaaa aactgagtgt gagagactgg caggcaccga gtctccagcg
60
agggaggagc cagaagagga cttccctgcg gcaccgcgct tatactggga cgacctgaag
120
agaaagttgt cagagaaact ggacaccaca gacttcacca gcaccatcaa gctgctgaat
180
gaaaatttat atgtccctcg tgaggctgga tctcaaaaag atgaaaatct tgcattgtat
240
attgaaaatc aatttcgtga atttaaacta agcaaagtct ggcgtgatca acattttgtt
300
aagattcagg tcaaggacag tgctcaaaac tcggtgatca tagttgataa gaatggtgga
360
cttgtttacc tggtggagaa tcctgggggt tatgtggcat atagtaaggc tgcaacagtt
420
actggtaaac tggtccatgc taattttggt actaaaaaag actttgagga tttagactct
480
cctgtgaatg gatctatagt gattgtcaga gcaggaaaaa tcacctttgc agaaaaggtt
540
gcaaatgctg aaagcttaaa tgcaattggt gtcttgatat atatggacca gactaaattt
600
cctattgtta aggcagacct ttcattcttt ggacatgctc atctgggaac aggtgaccct
660
tacacacctg gattcccttc cttcaatcac actcagtttc caccatctca gtcgtcagga
720
ttgcctaata tacctgtcca aacgatctcc agagctgctg cagaaaagct gtttggaaat
780
atggagggag actgtccctc tgactggaaa acagactcta cgtgtaagat ggtaacctcg
840
gaaaacaaga gtgtgaagct cacggtgagc aatgtgctga aagagacaaa aattcttaac
900
atctttggag ttattaaagg cttcgtagaa ccagatcact atgttgtggt tggggcccag
960
agagatgcgt ggggccccgg agctgcaaaa tccagtgtgg ggacagctct cctgttgaaa
1020
cttgcccaga tgttctcaga tatggtctta aaagatgggt ttcagcccag cagaagcatt
1080
atctttgcca gttggagtgc tggagacttt ggatcggttg gtgccactga atggctagag
1140
ggataccttt catccttgca tttaaaggct ttcacttaca ttaatctgga taaagcggtg
1200
cttggaacca gcaacttcaa ggtttctgcc agcccgttgt tgtatacgct gattgagaaa
1260
acaatgcaag atgtgaaaca tccggttact gggcgatctc tatatcagga cagcaactgg
1320
gccagcaaag ttgagaaact cactttagac aatgctgctt tccctttcct tgcgtattct
1380
ggaatcccag cagtttcttt ctgtttttgt gaggacacag attatcctta cttgggcacc
1440
accatggaca cctataagga actggtggag aggattcctg agctgaacaa agtggcacga
1500
gcagcggcag aagtagctgg tcagttcgtg attaaactga cccatgatac tgaattgaac
1560
ctggactatg agaggtacaa cagccagctg cttttgtttt tgagggatct gaaccagtac
1620
agagcagatg taaaggaaat gggcctgagc ttgcagtggc tgtattctgc tcgtggagac
1680
tttttccgtg ctacttccag gctaacaaca gatttcagga atgctgagaa aagggacaag
1740
tttgtcatga agaagctcaa tgatcgtgtc atgagagtcg agtattactt cctctcaccc
1800
tatgtgtctc caaaagagtc tcctttccgg cacgtcttct ggggctcggg ctcccacacg
1860
ctgtccgctt tactggagag cttgaaactg cgtagacaga ataacagtgc ttttaatgaa
1920
acgctgttca gaaaccagct ggctctcgcg acttggacta ttcagggagc tgcaaatgcc
1980
ctttctggtg acgtttggga cattgacaat gagttt
2016
<210> 4
<211> 672
<212> PRT
<213> Monkey
<400> 4
Cys Lys Gly Val Glu Pro Lys Thr Glu Cys Glu Arg Leu Ala Gly Thr
1 5 10 15
Glu Ser Pro Ala Arg Glu Glu Pro Glu Glu Asp Phe Pro Ala Ala Pro
20 25 30
Arg Leu Tyr Trp Asp Asp Leu Lys Arg Lys Leu Ser Glu Lys Leu Asp
35 40 45
Thr Thr Asp Phe Thr Ser Thr Ile Lys Leu Leu Asn Glu Asn Leu Tyr
50 55 60
Val Pro Arg Glu Ala Gly Ser Gln Lys Asp Glu Asn Leu Ala Leu Tyr
65 70 75 80
Ile Glu Asn Gln Phe Arg Glu Phe Lys Leu Ser Lys Val Trp Arg Asp
85 90 95
Gln His Phe Val Lys Ile Gln Val Lys Asp Ser Ala Gln Asn Ser Val
100 105 110
Ile Ile Val Asp Lys Asn Gly Gly Leu Val Tyr Leu Val Glu Asn Pro
115 120 125
Gly Gly Tyr Val Ala Tyr Ser Lys Ala Ala Thr Val Thr Gly Lys Leu
130 135 140
Val His Ala Asn Phe Gly Thr Lys Lys Asp Phe Glu Asp Leu Asp Ser
145 150 155 160
Pro Val Asn Gly Ser Ile Val Ile Val Arg Ala Gly Lys Ile Thr Phe
165 170 175
Ala Glu Lys Val Ala Asn Ala Glu Ser Leu Asn Ala Ile Gly Val Leu
180 185 190
Ile Tyr Met Asp Gln Thr Lys Phe Pro Ile Val Lys Ala Asp Leu Ser
195 200 205
Phe Phe Gly His Ala His Leu Gly Thr Gly Asp Pro Tyr Thr Pro Gly
210 215 220
Phe Pro Ser Phe Asn His Thr Gln Phe Pro Pro Ser Gln Ser Ser Gly
225 230 235 240
Leu Pro Asn Ile Pro Val Gln Thr Ile Ser Arg Ala Ala Ala Glu Lys
245 250 255
Leu Phe Gly Asn Met Glu Gly Asp Cys Pro Ser Asp Trp Lys Thr Asp
260 265 270
Ser Thr Cys Lys Met Val Thr Ser Glu Asn Lys Ser Val Lys Leu Thr
275 280 285
Val Ser Asn Val Leu Lys Glu Thr Lys Ile Leu Asn Ile Phe Gly Val
290 295 300
Ile Lys Gly Phe Val Glu Pro Asp His Tyr Val Val Val Gly Ala Gln
305 310 315 320
Arg Asp Ala Trp Gly Pro Gly Ala Ala Lys Ser Ser Val Gly Thr Ala
325 330 335
Leu Leu Leu Lys Leu Ala Gln Met Phe Ser Asp Met Val Leu Lys Asp
340 345 350
Gly Phe Gln Pro Ser Arg Ser Ile Ile Phe Ala Ser Trp Ser Ala Gly
355 360 365
Asp Phe Gly Ser Val Gly Ala Thr Glu Trp Leu Glu Gly Tyr Leu Ser
370 375 380
Ser Leu His Leu Lys Ala Phe Thr Tyr Ile Asn Leu Asp Lys Ala Val
385 390 395 400
Leu Gly Thr Ser Asn Phe Lys Val Ser Ala Ser Pro Leu Leu Tyr Thr
405 410 415
Leu Ile Glu Lys Thr Met Gln Asp Val Lys His Pro Val Thr Gly Arg
420 425 430
Ser Leu Tyr Gln Asp Ser Asn Trp Ala Ser Lys Val Glu Lys Leu Thr
435 440 445
Leu Asp Asn Ala Ala Phe Pro Phe Leu Ala Tyr Ser Gly Ile Pro Ala
450 455 460
Val Ser Phe Cys Phe Cys Glu Asp Thr Asp Tyr Pro Tyr Leu Gly Thr
465 470 475 480
Thr Met Asp Thr Tyr Lys Glu Leu Val Glu Arg Ile Pro Glu Leu Asn
485 490 495
Lys Val Ala Arg Ala Ala Ala Glu Val Ala Gly Gln Phe Val Ile Lys
500 505 510
Leu Thr His Asp Thr Glu Leu Asn Leu Asp Tyr Glu Arg Tyr Asn Ser
515 520 525
Gln Leu Leu Leu Phe Leu Arg Asp Leu Asn Gln Tyr Arg Ala Asp Val
530 535 540
Lys Glu Met Gly Leu Ser Leu Gln Trp Leu Tyr Ser Ala Arg Gly Asp
545 550 555 560
Phe Phe Arg Ala Thr Ser Arg Leu Thr Thr Asp Phe Arg Asn Ala Glu
565 570 575
Lys Arg Asp Lys Phe Val Met Lys Lys Leu Asn Asp Arg Val Met Arg
580 585 590
Val Glu Tyr Tyr Phe Leu Ser Pro Tyr Val Ser Pro Lys Glu Ser Pro
595 600 605
Phe Arg His Val Phe Trp Gly Ser Gly Ser His Thr Leu Ser Ala Leu
610 615 620
Leu Glu Ser Leu Lys Leu Arg Arg Gln Asn Asn Ser Ala Phe Asn Glu
625 630 635 640
Thr Leu Phe Arg Asn Gln Leu Ala Leu Ala Thr Trp Thr Ile Gln Gly
645 650 655
Ala Ala Asn Ala Leu Ser Gly Asp Val Trp Asp Ile Asp Asn Glu Phe
660 665 670
<210> 5
<211> 2280
<212> DNA
<213> Homo sapiens
<400> 5
atgatggatc aagctagatc agcattctct aacttgtttg gtggagaacc attgtcatat
60
acccggttca gcctggctcg gcaagtagat ggcgataaca gtcatgtgga gatgaaactt
120
gctgtagatg aagaagaaaa tgctgacaat aacacaaagg ccaatgtcac aaaaccaaaa
180
aggtgtagtg gaagtatctg ctatgggact attgctgtga tcgtcttttt cttgattgga
240
tttatgattg gctacttggg ctattgtaaa ggggtagaac caaaaactga gtgtgagaga
300
ctggcaggaa ccgagtctcc agtgagggag gagccaggag aggacttccc tgcagcacgt
360
cgcttatatt gggatgacct gaagagaaag ttgtcggaga aactggacag cacagacttc
420
accggcacca tcaagctgct gaatgaaaat tcatatgtcc ctcgtgaggc tggatctcaa
480
aaagatgaaa atcttgcgtt gtatgttgaa aatcaatttc gtgaatttaa actcagcaaa
540
gtctggcgtg atcaacattt tgttaagatt caggtcaaag acagcgctca aaactcggtg
600
atcatagttg ataagaacgg tagacttgtt tacctggtgg agaatcctgg gggttatgtg
660
gcgtatagta aggctgcaac agttactggt aaactggtcc atgctaattt tggtactaaa
720
aaagattttg aggatttata cactcctgtg aatggatcta tagtgattgt cagagcaggg
780
aaaatcacct ttgcagaaaa ggttgcaaat gctgaaagct taaatgcaat tggtgtgttg
840
atatacatgg accagactaa atttcccatt gttaacgcag aactttcatt ctttggacat
900
gctcatctgg ggacaggtga cccttacaca cctggattcc cttccttcaa tcacactcag
960
tttccaccat ctcggtcatc aggattgcct aatatacctg tccagacaat ctccagagct
1020
gctgcagaaa agctgtttgg gaatatggaa ggagactgtc cctctgactg gaaaacagac
1080
tctacatgta ggatggtaac ctcagaaagc aagaatgtga agctcactgt gagcaatgtg
1140
ctgaaagaga taaaaattct taacatcttt ggagttatta aaggctttgt agaaccagat
1200
cactatgttg tagttggggc ccagagagat gcatggggcc ctggagctgc aaaatccggt
1260
gtaggcacag ctctcctatt gaaacttgcc cagatgttct cagatatggt cttaaaagat
1320
gggtttcagc ccagcagaag cattatcttt gccagttgga gtgctggaga ctttggatcg
1380
gttggtgcca ctgaatggct agagggatac ctttcgtccc tgcatttaaa ggctttcact
1440
tatattaatc tggataaagc ggttcttggt accagcaact tcaaggtttc tgccagccca
1500
ctgttgtata cgcttattga gaaaacaatg caaaatgtga agcatccggt tactgggcaa
1560
tttctatatc aggacagcaa ctgggccagc aaagttgaga aactcacttt agacaatgct
1620
gctttccctt tccttgcata ttctggaatc ccagcagttt ctttctgttt ttgcgaggac
1680
acagattatc cttatttggg taccaccatg gacacctata aggaactgat tgagaggatt
1740
cctgagttga acaaagtggc acgagcagct gcagaggtcg ctggtcagtt cgtgattaaa
1800
ctaacccatg atgttgaatt gaacctggac tatgagaggt acaacagcca actgctttca
1860
tttgtgaggg atctgaacca atacagagca gacataaagg aaatgggcct gagtttacag
1920
tggctgtatt ctgctcgtgg agacttcttc cgtgctactt ccagactaac aacagatttc
1980
gggaatgctg agaaaacaga cagatttgtc atgaagaaac tcaatgatcg tgtcatgaga
2040
gtggagtatc acttcctctc tccctacgta tctccaaaag agtctccttt ccgacatgtc
2100
ttctggggct ccggctctca cacgctgcca gctttactgg agaacttgaa actgcgtaaa
2160
caaaataacg gtgcttttaa tgaaacgctg ttcagaaacc agttggctct agctacttgg
2220
actattcagg gagctgcaaa tgccctctct ggtgacgttt gggacattga caatgagttt
2280
<210> 6
<211> 760
<212> PRT
<213> Homo sapiens
<400> 6
Met Met Asp Gln Ala Arg Ser Ala Phe Ser Asn Leu Phe Gly Gly Glu
1 5 10 15
Pro Leu Ser Tyr Thr Arg Phe Ser Leu Ala Arg Gln Val Asp Gly Asp
20 25 30
Asn Ser His Val Glu Met Lys Leu Ala Val Asp Glu Glu Glu Asn Ala
35 40 45
Asp Asn Asn Thr Lys Ala Asn Val Thr Lys Pro Lys Arg Cys Ser Gly
50 55 60
Ser Ile Cys Tyr Gly Thr Ile Ala Val Ile Val Phe Phe Leu Ile Gly
65 70 75 80
Phe Met Ile Gly Tyr Leu Gly Tyr Cys Lys Gly Val Glu Pro Lys Thr
85 90 95
Glu Cys Glu Arg Leu Ala Gly Thr Glu Ser Pro Val Arg Glu Glu Pro
100 105 110
Gly Glu Asp Phe Pro Ala Ala Arg Arg Leu Tyr Trp Asp Asp Leu Lys
115 120 125
Arg Lys Leu Ser Glu Lys Leu Asp Ser Thr Asp Phe Thr Gly Thr Ile
130 135 140
Lys Leu Leu Asn Glu Asn Ser Tyr Val Pro Arg Glu Ala Gly Ser Gln
145 150 155 160
Lys Asp Glu Asn Leu Ala Leu Tyr Val Glu Asn Gln Phe Arg Glu Phe
165 170 175
Lys Leu Ser Lys Val Trp Arg Asp Gln His Phe Val Lys Ile Gln Val
180 185 190
Lys Asp Ser Ala Gln Asn Ser Val Ile Ile Val Asp Lys Asn Gly Arg
195 200 205
Leu Val Tyr Leu Val Glu Asn Pro Gly Gly Tyr Val Ala Tyr Ser Lys
210 215 220
Ala Ala Thr Val Thr Gly Lys Leu Val His Ala Asn Phe Gly Thr Lys
225 230 235 240
Lys Asp Phe Glu Asp Leu Tyr Thr Pro Val Asn Gly Ser Ile Val Ile
245 250 255
Val Arg Ala Gly Lys Ile Thr Phe Ala Glu Lys Val Ala Asn Ala Glu
260 265 270
Ser Leu Asn Ala Ile Gly Val Leu Ile Tyr Met Asp Gln Thr Lys Phe
275 280 285
Pro Ile Val Asn Ala Glu Leu Ser Phe Phe Gly His Ala His Leu Gly
290 295 300
Thr Gly Asp Pro Tyr Thr Pro Gly Phe Pro Ser Phe Asn His Thr Gln
305 310 315 320
Phe Pro Pro Ser Arg Ser Ser Gly Leu Pro Asn Ile Pro Val Gln Thr
325 330 335
Ile Ser Arg Ala Ala Ala Glu Lys Leu Phe Gly Asn Met Glu Gly Asp
340 345 350
Cys Pro Ser Asp Trp Lys Thr Asp Ser Thr Cys Arg Met Val Thr Ser
355 360 365
Glu Ser Lys Asn Val Lys Leu Thr Val Ser Asn Val Leu Lys Glu Ile
370 375 380
Lys Ile Leu Asn Ile Phe Gly Val Ile Lys Gly Phe Val Glu Pro Asp
385 390 395 400
His Tyr Val Val Val Gly Ala Gln Arg Asp Ala Trp Gly Pro Gly Ala
405 410 415
Ala Lys Ser Gly Val Gly Thr Ala Leu Leu Leu Lys Leu Ala Gln Met
420 425 430
Phe Ser Asp Met Val Leu Lys Asp Gly Phe Gln Pro Ser Arg Ser Ile
435 440 445
Ile Phe Ala Ser Trp Ser Ala Gly Asp Phe Gly Ser Val Gly Ala Thr
450 455 460
Glu Trp Leu Glu Gly Tyr Leu Ser Ser Leu His Leu Lys Ala Phe Thr
465 470 475 480
Tyr Ile Asn Leu Asp Lys Ala Val Leu Gly Thr Ser Asn Phe Lys Val
485 490 495
Ser Ala Ser Pro Leu Leu Tyr Thr Leu Ile Glu Lys Thr Met Gln Asn
500 505 510
Val Lys His Pro Val Thr Gly Gln Phe Leu Tyr Gln Asp Ser Asn Trp
515 520 525
Ala Ser Lys Val Glu Lys Leu Thr Leu Asp Asn Ala Ala Phe Pro Phe
530 535 540
Leu Ala Tyr Ser Gly Ile Pro Ala Val Ser Phe Cys Phe Cys Glu Asp
545 550 555 560
Thr Asp Tyr Pro Tyr Leu Gly Thr Thr Met Asp Thr Tyr Lys Glu Leu
565 570 575
Ile Glu Arg Ile Pro Glu Leu Asn Lys Val Ala Arg Ala Ala Ala Glu
580 585 590
Val Ala Gly Gln Phe Val Ile Lys Leu Thr His Asp Val Glu Leu Asn
595 600 605
Leu Asp Tyr Glu Arg Tyr Asn Ser Gln Leu Leu Ser Phe Val Arg Asp
610 615 620
Leu Asn Gln Tyr Arg Ala Asp Ile Lys Glu Met Gly Leu Ser Leu Gln
625 630 635 640
Trp Leu Tyr Ser Ala Arg Gly Asp Phe Phe Arg Ala Thr Ser Arg Leu
645 650 655
Thr Thr Asp Phe Gly Asn Ala Glu Lys Thr Asp Arg Phe Val Met Lys
660 665 670
Lys Leu Asn Asp Arg Val Met Arg Val Glu Tyr His Phe Leu Ser Pro
675 680 685
Tyr Val Ser Pro Lys Glu Ser Pro Phe Arg His Val Phe Trp Gly Ser
690 695 700
Gly Ser His Thr Leu Pro Ala Leu Leu Glu Asn Leu Lys Leu Arg Lys
705 710 715 720
Gln Asn Asn Gly Ala Phe Asn Glu Thr Leu Phe Arg Asn Gln Leu Ala
725 730 735
Leu Ala Thr Trp Thr Ile Gln Gly Ala Ala Asn Ala Leu Ser Gly Asp
740 745 750
Val Trp Asp Ile Asp Asn Glu Phe
755 760
<210> 7
<211> 2280
<212> DNA
<213> Cynomolgus monkey
<400> 7
atgatggatc aagctagatc agcattctct aacttgtttg gtggagaacc attgtcatat
60
acccggttca gcctggctcg gcaagtagac ggcgataaca gtcatgtgga gatgaaactt
120
gctgtagatg atgaagaaaa tgctgacaat aacacaaagg ccaatggcac aaaaccaaaa
180
aggtgtggtg gaaatatctg ctatgggact attgccgtga tcatcttttt cttgattggg
240
tttatgattg gctacttggg ctattgtaaa ggggtagaac caaaaactga gtgtgagaga
300
ctggcaggca ccgagtctcc agcgagggag gagccagaag aggacttccc tgcggcaccg
360
cgcttatact gggacgacct gaagagaaag ttgtcagaga aactggacac cacagacttc
420
accagcacca tcaagctgct gaatgaaaat ttatatgtcc ctcgtgaggc tggatctcaa
480
aaagatgaaa atcttgcatt gtatattgaa aatcaatttc gtgaatttaa actaagcaaa
540
gtctggcgtg atcaacattt tgttaagatt caggtcaagg acagtgctca aaactcggtg
600
atcatagttg ataagaatgg tggacttgtt tacctggtgg agaatcctgg gggttatgtg
660
gcatatagta aggctgcaac agttactggt aaactggtcc atgctaattt tggtactaaa
720
aaagactttg aggatttaga ctctcctgtg aatggatcta tagtgattgt cagagcagga
780
aaaatcacct ttgcagaaaa ggttgcaaat gctgaaagct taaatgcaat tggtgtcttg
840
atatatatgg accagactaa atttcctatt gttaaggcag acctttcatt ctttggacat
900
gctcatctgg gaacaggtga cccttacaca cctggattcc cttccttcaa tcacactcag
960
tttccaccat ctcagtcgtc aggattgcct aatatacctg tccaaacgat ctccagagct
1020
gctgcagaaa agctgtttgg aaatatggag ggagactgtc cctctgactg gaaaacagac
1080
tctacgtgta agatggtaac ctcggaaaac aagagtgtga agctcacggt gagcaatgtg
1140
ctgaaagaga caaaaattct taacatcttt ggagttatta aaggcttcgt agaaccagat
1200
cactatgttg tggttggggc ccagagagat gcgtggggcc ccggagctgc aaaatccagt
1260
gtggggacag ctctcctgtt gaaacttgcc cagatgttct cagatatggt cttaaaagat
1320
gggtttcagc ccagcagaag cattatcttt gccagttgga gtgctggaga ctttggatcg
1380
gttggtgcca ctgaatggct agagggatac ctttcatcct tgcatttaaa ggctttcact
1440
tacattaatc tggataaagc ggtgcttgga accagcaact tcaaggtttc tgccagcccg
1500
ttgttgtata cgctgattga gaaaacaatg caagatgtga aacatccggt tactgggcga
1560
tctctatatc aggacagcaa ctgggccagc aaagttgaga aactcacttt agacaatgct
1620
gctttccctt tccttgcgta ttctggaatc ccagcagttt ctttctgttt ttgtgaggac
1680
acagattatc cttacttggg caccaccatg gacacctata aggaactggt ggagaggatt
1740
cctgagctga acaaagtggc acgagcagcg gcagaagtag ctggtcagtt cgtgattaaa
1800
ctgacccatg atactgaatt gaacctggac tatgagaggt acaacagcca gctgcttttg
1860
tttttgaggg atctgaacca gtacagagca gatgtaaagg aaatgggcct gagcttgcag
1920
tggctgtatt ctgctcgtgg agactttttc cgtgctactt ccaggctaac aacagatttc
1980
aggaatgctg agaaaaggga caagtttgtc atgaagaagc tcaatgatcg tgtcatgaga
2040
gtcgagtatt acttcctctc accctatgtg tctccaaaag agtctccttt ccggcacgtc
2100
ttctggggct cgggctccca cacgctgtcc gctttactgg agagcttgaa actgcgtaga
2160
cagaataaca gtgcttttaa tgaaacgctg ttcagaaacc agctggctct cgcgacttgg
2220
actattcagg gagctgcaaa tgccctttct ggtgacgttt gggacattga caatgagttt
2280
<210> 8
<211> 760
<212> PRT
<213> Cynomolgus monkey
<400> 8
Met Met Asp Gln Ala Arg Ser Ala Phe Ser Asn Leu Phe Gly Gly Glu
1 5 10 15
Pro Leu Ser Tyr Thr Arg Phe Ser Leu Ala Arg Gln Val Asp Gly Asp
20 25 30
Asn Ser His Val Glu Met Lys Leu Ala Val Asp Asp Glu Glu Asn Ala
35 40 45
Asp Asn Asn Thr Lys Ala Asn Gly Thr Lys Pro Lys Arg Cys Gly Gly
50 55 60
Asn Ile Cys Tyr Gly Thr Ile Ala Val Ile Ile Phe Phe Leu Ile Gly
65 70 75 80
Phe Met Ile Gly Tyr Leu Gly Tyr Cys Lys Gly Val Glu Pro Lys Thr
85 90 95
Glu Cys Glu Arg Leu Ala Gly Thr Glu Ser Pro Ala Arg Glu Glu Pro
100 105 110
Glu Glu Asp Phe Pro Ala Ala Pro Arg Leu Tyr Trp Asp Asp Leu Lys
115 120 125
Arg Lys Leu Ser Glu Lys Leu Asp Thr Thr Asp Phe Thr Ser Thr Ile
130 135 140
Lys Leu Leu Asn Glu Asn Leu Tyr Val Pro Arg Glu Ala Gly Ser Gln
145 150 155 160
Lys Asp Glu Asn Leu Ala Leu Tyr Ile Glu Asn Gln Phe Arg Glu Phe
165 170 175
Lys Leu Ser Lys Val Trp Arg Asp Gln His Phe Val Lys Ile Gln Val
180 185 190
Lys Asp Ser Ala Gln Asn Ser Val Ile Ile Val Asp Lys Asn Gly Gly
195 200 205
Leu Val Tyr Leu Val Glu Asn Pro Gly Gly Tyr Val Ala Tyr Ser Lys
210 215 220
Ala Ala Thr Val Thr Gly Lys Leu Val His Ala Asn Phe Gly Thr Lys
225 230 235 240
Lys Asp Phe Glu Asp Leu Asp Ser Pro Val Asn Gly Ser Ile Val Ile
245 250 255
Val Arg Ala Gly Lys Ile Thr Phe Ala Glu Lys Val Ala Asn Ala Glu
260 265 270
Ser Leu Asn Ala Ile Gly Val Leu Ile Tyr Met Asp Gln Thr Lys Phe
275 280 285
Pro Ile Val Lys Ala Asp Leu Ser Phe Phe Gly His Ala His Leu Gly
290 295 300
Thr Gly Asp Pro Tyr Thr Pro Gly Phe Pro Ser Phe Asn His Thr Gln
305 310 315 320
Phe Pro Pro Ser Gln Ser Ser Gly Leu Pro Asn Ile Pro Val Gln Thr
325 330 335
Ile Ser Arg Ala Ala Ala Glu Lys Leu Phe Gly Asn Met Glu Gly Asp
340 345 350
Cys Pro Ser Asp Trp Lys Thr Asp Ser Thr Cys Lys Met Val Thr Ser
355 360 365
Glu Asn Lys Ser Val Lys Leu Thr Val Ser Asn Val Leu Lys Glu Thr
370 375 380
Lys Ile Leu Asn Ile Phe Gly Val Ile Lys Gly Phe Val Glu Pro Asp
385 390 395 400
His Tyr Val Val Val Gly Ala Gln Arg Asp Ala Trp Gly Pro Gly Ala
405 410 415
Ala Lys Ser Ser Val Gly Thr Ala Leu Leu Leu Lys Leu Ala Gln Met
420 425 430
Phe Ser Asp Met Val Leu Lys Asp Gly Phe Gln Pro Ser Arg Ser Ile
435 440 445
Ile Phe Ala Ser Trp Ser Ala Gly Asp Phe Gly Ser Val Gly Ala Thr
450 455 460
Glu Trp Leu Glu Gly Tyr Leu Ser Ser Leu His Leu Lys Ala Phe Thr
465 470 475 480
Tyr Ile Asn Leu Asp Lys Ala Val Leu Gly Thr Ser Asn Phe Lys Val
485 490 495
Ser Ala Ser Pro Leu Leu Tyr Thr Leu Ile Glu Lys Thr Met Gln Asp
500 505 510
Val Lys His Pro Val Thr Gly Arg Ser Leu Tyr Gln Asp Ser Asn Trp
515 520 525
Ala Ser Lys Val Glu Lys Leu Thr Leu Asp Asn Ala Ala Phe Pro Phe
530 535 540
Leu Ala Tyr Ser Gly Ile Pro Ala Val Ser Phe Cys Phe Cys Glu Asp
545 550 555 560
Thr Asp Tyr Pro Tyr Leu Gly Thr Thr Met Asp Thr Tyr Lys Glu Leu
565 570 575
Val Glu Arg Ile Pro Glu Leu Asn Lys Val Ala Arg Ala Ala Ala Glu
580 585 590
Val Ala Gly Gln Phe Val Ile Lys Leu Thr His Asp Thr Glu Leu Asn
595 600 605
Leu Asp Tyr Glu Arg Tyr Asn Ser Gln Leu Leu Leu Phe Leu Arg Asp
610 615 620
Leu Asn Gln Tyr Arg Ala Asp Val Lys Glu Met Gly Leu Ser Leu Gln
625 630 635 640
Trp Leu Tyr Ser Ala Arg Gly Asp Phe Phe Arg Ala Thr Ser Arg Leu
645 650 655
Thr Thr Asp Phe Arg Asn Ala Glu Lys Arg Asp Lys Phe Val Met Lys
660 665 670
Lys Leu Asn Asp Arg Val Met Arg Val Glu Tyr Tyr Phe Leu Ser Pro
675 680 685
Tyr Val Ser Pro Lys Glu Ser Pro Phe Arg His Val Phe Trp Gly Ser
690 695 700
Gly Ser His Thr Leu Ser Ala Leu Leu Glu Ser Leu Lys Leu Arg Arg
705 710 715 720
Gln Asn Asn Ser Ala Phe Asn Glu Thr Leu Phe Arg Asn Gln Leu Ala
725 730 735
Leu Ala Thr Trp Thr Ile Gln Gly Ala Ala Asn Ala Leu Ser Gly Asp
740 745 750
Val Trp Asp Ile Asp Asn Glu Phe
755 760
<210> 9
<211> 1914
<212> DNA
<213> Homo sapiens
<400> 9
atgcgaccct ccgggacggc cggggcagcg ctcctggcgc tgctggctgc gctctgcccg
60
gcgagtcggg ctctggagga aaagaaagtt tgccaaggca cgagtaacaa gctcacgcag
120
ttgggcactt ttgaagatca ttttctcagc ctccagagga tgttcaataa ctgtgaggtg
180
gtccttggga atttggaaat tacctatgtg cagaggaatt atgatctttc cttcttaaag
240
accatccagg aggtggctgg ttatgtcctc attgccctca acacagtgga gcgaattcct
300
ttggaaaacc tgcagatcat cagaggaaat atgtactacg aaaattccta tgccttagca
360
gtcttatcta actatgatgc aaataaaacc ggactgaagg agctgcccat gagaaattta
420
caggaaatcc tgcatggcgc cgtgcggttc agcaacaacc ctgccctgtg caacgtggag
480
agcatccagt ggcgggacat agtcagcagt gactttctca gcaacatgtc gatggacttc
540
cagaaccacc tgggcagctg ccaaaagtgt gatccaagct gtcccaatgg gagctgctgg
600
ggtgcaggag aggagaactg ccagaaactg accaaaatca tctgtgccca gcagtgctcc
660
gggcgctgcc gtggcaagtc ccccagtgac tgctgccaca accagtgtgc tgcaggctgc
720
acaggccccc gggagagcga ctgcctggtc tgccgcaaat tccgagacga agccacgtgc
780
aaggacacct gccccccact catgctctac aaccccacca cgtaccagat ggatgtgaac
840
cccgagggca aatacagctt tggtgccacc tgcgtgaaga agtgtccccg taattatgtg
900
gtgacagatc acggctcgtg cgtccgagcc tgtggggccg acagctatga gatggaggaa
960
gacggcgtcc gcaagtgtaa gaagtgcgaa gggccttgcc gcaaagtgtg taacggaata
1020
ggtattggtg aatttaaaga ctcactctcc ataaatgcta cgaatattaa acacttcaaa
1080
aactgcacct ccatcagtgg cgatctccac atcctgccgg tggcatttag gggtgactcc
1140
ttcacacata ctcctcctct ggatccacag gaactggata ttctgaaaac cgtaaaggaa
1200
atcacagggt ttttgctgat tcaggcttgg cctgaaaaca ggacggacct ccatgccttt
1260
gagaacctag aaatcatacg cggcaggacc aagcaacatg gtcagttttc tcttgcagtc
1320
gtcagcctga acataacatc cttgggatta cgctccctca aggagataag tgatggagat
1380
gtgataattt caggaaacaa aaatttgtgc tatgcaaata caataaactg gaaaaaactg
1440
tttgggacct ccggtcagaa aaccaaaatt ataagcaaca gaggtgaaaa cagctgcaag
1500
gccacaggcc aggtctgcca tgccttgtgc tcccccgagg gctgctgggg cccggagccc
1560
agggactgcg tctcttgccg gaatgtcagc cgaggcaggg aatgcgtgga caagtgcaac
1620
cttctggagg gtgagccaag ggagtttgtg gagaactctg agtgcataca gtgccaccca
1680
gagtgcctgc ctcaggccat gaacatcacc tgcacaggac ggggaccaga caactgtatc
1740
cagtgtgccc actacattga cggcccccac tgcgtcaaga cctgcccggc aggagtcatg
1800
ggagaaaaca acaccctggt ctggaagtac gcagacgccg gccatgtgtg ccacctgtgc
1860
catccaaact gcacctacgg atgcactggg ccaggtcttg aaggctgtcc aacg
1914
<210> 10
<211> 638
<212> PRT
<213> Homo sapiens
<400> 10
Met Arg Pro Ser Gly Thr Ala Gly Ala Ala Leu Leu Ala Leu Leu Ala
1 5 10 15
Ala Leu Cys Pro Ala Ser Arg Ala Leu Glu Glu Lys Lys Val Cys Gln
20 25 30
Gly Thr Ser Asn Lys Leu Thr Gln Leu Gly Thr Phe Glu Asp His Phe
35 40 45
Leu Ser Leu Gln Arg Met Phe Asn Asn Cys Glu Val Val Leu Gly Asn
50 55 60
Leu Glu Ile Thr Tyr Val Gln Arg Asn Tyr Asp Leu Ser Phe Leu Lys
65 70 75 80
Thr Ile Gln Glu Val Ala Gly Tyr Val Leu Ile Ala Leu Asn Thr Val
85 90 95
Glu Arg Ile Pro Leu Glu Asn Leu Gln Ile Ile Arg Gly Asn Met Tyr
100 105 110
Tyr Glu Asn Ser Tyr Ala Leu Ala Val Leu Ser Asn Tyr Asp Ala Asn
115 120 125
Lys Thr Gly Leu Lys Glu Leu Pro Met Arg Asn Leu Gln Glu Ile Leu
130 135 140
His Gly Ala Val Arg Phe Ser Asn Asn Pro Ala Leu Cys Asn Val Glu
145 150 155 160
Ser Ile Gln Trp Arg Asp Ile Val Ser Ser Asp Phe Leu Ser Asn Met
165 170 175
Ser Met Asp Phe Gln Asn His Leu Gly Ser Cys Gln Lys Cys Asp Pro
180 185 190
Ser Cys Pro Asn Gly Ser Cys Trp Gly Ala Gly Glu Glu Asn Cys Gln
195 200 205
Lys Leu Thr Lys Ile Ile Cys Ala Gln Gln Cys Ser Gly Arg Cys Arg
210 215 220
Gly Lys Ser Pro Ser Asp Cys Cys His Asn Gln Cys Ala Ala Gly Cys
225 230 235 240
Thr Gly Pro Arg Glu Ser Asp Cys Leu Val Cys Arg Lys Phe Arg Asp
245 250 255
Glu Ala Thr Cys Lys Asp Thr Cys Pro Pro Leu Met Leu Tyr Asn Pro
260 265 270
Thr Thr Tyr Gln Met Asp Val Asn Pro Glu Gly Lys Tyr Ser Phe Gly
275 280 285
Ala Thr Cys Val Lys Lys Cys Pro Arg Asn Tyr Val Val Thr Asp His
290 295 300
Gly Ser Cys Val Arg Ala Cys Gly Ala Asp Ser Tyr Glu Met Glu Glu
305 310 315 320
Asp Gly Val Arg Lys Cys Lys Lys Cys Glu Gly Pro Cys Arg Lys Val
325 330 335
Cys Asn Gly Ile Gly Ile Gly Glu Phe Lys Asp Ser Leu Ser Ile Asn
340 345 350
Ala Thr Asn Ile Lys His Phe Lys Asn Cys Thr Ser Ile Ser Gly Asp
355 360 365
Leu His Ile Leu Pro Val Ala Phe Arg Gly Asp Ser Phe Thr His Thr
370 375 380
Pro Pro Leu Asp Pro Gln Glu Leu Asp Ile Leu Lys Thr Val Lys Glu
385 390 395 400
Ile Thr Gly Phe Leu Leu Ile Gln Ala Trp Pro Glu Asn Arg Thr Asp
405 410 415
Leu His Ala Phe Glu Asn Leu Glu Ile Ile Arg Gly Arg Thr Lys Gln
420 425 430
His Gly Gln Phe Ser Leu Ala Val Val Ser Leu Asn Ile Thr Ser Leu
435 440 445
Gly Leu Arg Ser Leu Lys Glu Ile Ser Asp Gly Asp Val Ile Ile Ser
450 455 460
Gly Asn Lys Asn Leu Cys Tyr Ala Asn Thr Ile Asn Trp Lys Lys Leu
465 470 475 480
Phe Gly Thr Ser Gly Gln Lys Thr Lys Ile Ile Ser Asn Arg Gly Glu
485 490 495
Asn Ser Cys Lys Ala Thr Gly Gln Val Cys His Ala Leu Cys Ser Pro
500 505 510
Glu Gly Cys Trp Gly Pro Glu Pro Arg Asp Cys Val Ser Cys Arg Asn
515 520 525
Val Ser Arg Gly Arg Glu Cys Val Asp Lys Cys Asn Leu Leu Glu Gly
530 535 540
Glu Pro Arg Glu Phe Val Glu Asn Ser Glu Cys Ile Gln Cys His Pro
545 550 555 560
Glu Cys Leu Pro Gln Ala Met Asn Ile Thr Cys Thr Gly Arg Gly Pro
565 570 575
Asp Asn Cys Ile Gln Cys Ala His Tyr Ile Asp Gly Pro His Cys Val
580 585 590
Lys Thr Cys Pro Ala Gly Val Met Gly Glu Asn Asn Thr Leu Val Trp
595 600 605
Lys Tyr Ala Asp Ala Gly His Val Cys His Leu Cys His Pro Asn Cys
610 615 620
Thr Tyr Gly Cys Thr Gly Pro Gly Leu Glu Gly Cys Pro Thr
625 630 635
<210> 11
<211> 1914
<212> DNA
<213> Monkey
<400> 11
atgcgaccct ccgggacggc cggggccgcg ctcctggcgc tgctggctgc gctctgcccc
60
gcgagtcggg ctctggagga aaagaaagtt tgccaaggca cgagtaacaa actcacgcag
120
ttgggcactt ttgaagatca ttttctcagc ctccagagga tgttcaataa ctgtgaggtg
180
gtccttggga atttggaaat tacctacgtg cagaggaatt atgatctttc cttcttaaag
240
accatccagg aggtggctgg ttatgtcctc atcgccctca acacagtgga gcggattcct
300
ttggaaaacc tgcagatcat cagaggaaac atgtactatg aaaattccta tgccttagca
360
gtcttatcta actatgatgc aaataaaacc ggactgaagg agctgcccat gagaaactta
420
caggaaatcc tgcatggcgc cgtgcggttc agcaacaacc ctgccctgtg caacgtggag
480
agcatccagt ggcgggacat agtcagcagc gagtttctca gcaacatgtc gatggacttc
540
cagaaccacc tgggcagctg ccaaaagtgt gatccaagct gtcccaatgg gagctgctgg
600
ggtgcaggag aggagaactg ccagaaactg accaaaatca tctgtgccca gcagtgctcc
660
gggcgctgcc gcggcaagtc ccccagtgac tgctgccaca accagtgtgc cgcgggctgc
720
acgggccccc gggagagcga ctgcctggtc tgccgcaaat tccgagacga agccacgtgc
780
aaggacacct gccccccact catgctctac aaccccacca cataccagat ggatgtgaac
840
cccgagggca aatacagctt tggtgccacc tgcgtgaaga agtgtccccg taattatgtg
900
gtgacagatc acggctcgtg cgtccgagcc tgcggggccg acagctatga gatggaggaa
960
gacggcgtcc gcaagtgtaa gaagtgcgaa gggccttgcc gcaaagtgtg taatggaata
1020
ggtattggtg aatttaaaga cacactctcc ataaatgcta caaatattaa acacttcaaa
1080
aactgcacct ccatcagtgg cgatctccac atcctgccgg tggcatttag gggtgactcc
1140
ttcacacaca ctccgcctct ggatccacag gaactggata ttctgaaaac cgtaaaggaa
1200
atcacagggt ttttgctgat tcaggcttgg cctgaaaaca ggacggacct ccatgctttt
1260
gagaacctag aaatcatacg tggcaggacc aagcaacacg gtcagttttc tcttgcggtc
1320
gtcagcctga acataacatc cttgggatta cgctccctca aggagataag cgatggagat
1380
gtgataattt caggaaacaa aaatttgtgc tatgcaaata caataaactg gaaaaaactg
1440
tttgggacct ccagtcagaa aaccaaaatt ataagcaaca gaggtgaaaa cagctgcaag
1500
gccacgggcc aggtctgcca tgccttgtgc tcccccgagg gctgctgggg cccggagccc
1560
agggactgcg tctcctgtca gaatgtcagc cgaggcagag aatgcgtgga caagtgcaac
1620
atcctggagg gcgagccaag ggagtttgtg gagaactctg agtgcataca gtgccaccca
1680
gaatgcctgc cccaggtcat gaacatcacc tgcacaggac ggggaccaga caactgtatc
1740
cagtgtgccc actacattga cggcccccac tgcgtcaaga cctgcccagc aggagtcatg
1800
ggagaaaaca acaccctggt ctggaagtac gcagacgccg gccacgtgtg ccacttgtgc
1860
catccaaact gcacctacgg atgcactggg ccaggtcttg aaggctgtgc aagg
1914
<210> 12
<211> 638
<212> PRT
<213> Monkey
<400> 12
Met Arg Pro Ser Gly Thr Ala Gly Ala Ala Leu Leu Ala Leu Leu Ala
1 5 10 15
Ala Leu Cys Pro Ala Ser Arg Ala Leu Glu Glu Lys Lys Val Cys Gln
20 25 30
Gly Thr Ser Asn Lys Leu Thr Gln Leu Gly Thr Phe Glu Asp His Phe
35 40 45
Leu Ser Leu Gln Arg Met Phe Asn Asn Cys Glu Val Val Leu Gly Asn
50 55 60
Leu Glu Ile Thr Tyr Val Gln Arg Asn Tyr Asp Leu Ser Phe Leu Lys
65 70 75 80
Thr Ile Gln Glu Val Ala Gly Tyr Val Leu Ile Ala Leu Asn Thr Val
85 90 95
Glu Arg Ile Pro Leu Glu Asn Leu Gln Ile Ile Arg Gly Asn Met Tyr
100 105 110
Tyr Glu Asn Ser Tyr Ala Leu Ala Val Leu Ser Asn Tyr Asp Ala Asn
115 120 125
Lys Thr Gly Leu Lys Glu Leu Pro Met Arg Asn Leu Gln Glu Ile Leu
130 135 140
His Gly Ala Val Arg Phe Ser Asn Asn Pro Ala Leu Cys Asn Val Glu
145 150 155 160
Ser Ile Gln Trp Arg Asp Ile Val Ser Ser Glu Phe Leu Ser Asn Met
165 170 175
Ser Met Asp Phe Gln Asn His Leu Gly Ser Cys Gln Lys Cys Asp Pro
180 185 190
Ser Cys Pro Asn Gly Ser Cys Trp Gly Ala Gly Glu Glu Asn Cys Gln
195 200 205
Lys Leu Thr Lys Ile Ile Cys Ala Gln Gln Cys Ser Gly Arg Cys Arg
210 215 220
Gly Lys Ser Pro Ser Asp Cys Cys His Asn Gln Cys Ala Ala Gly Cys
225 230 235 240
Thr Gly Pro Arg Glu Ser Asp Cys Leu Val Cys Arg Lys Phe Arg Asp
245 250 255
Glu Ala Thr Cys Lys Asp Thr Cys Pro Pro Leu Met Leu Tyr Asn Pro
260 265 270
Thr Thr Tyr Gln Met Asp Val Asn Pro Glu Gly Lys Tyr Ser Phe Gly
275 280 285
Ala Thr Cys Val Lys Lys Cys Pro Arg Asn Tyr Val Val Thr Asp His
290 295 300
Gly Ser Cys Val Arg Ala Cys Gly Ala Asp Ser Tyr Glu Met Glu Glu
305 310 315 320
Asp Gly Val Arg Lys Cys Lys Lys Cys Glu Gly Pro Cys Arg Lys Val
325 330 335
Cys Asn Gly Ile Gly Ile Gly Glu Phe Lys Asp Thr Leu Ser Ile Asn
340 345 350
Ala Thr Asn Ile Lys His Phe Lys Asn Cys Thr Ser Ile Ser Gly Asp
355 360 365
Leu His Ile Leu Pro Val Ala Phe Arg Gly Asp Ser Phe Thr His Thr
370 375 380
Pro Pro Leu Asp Pro Gln Glu Leu Asp Ile Leu Lys Thr Val Lys Glu
385 390 395 400
Ile Thr Gly Phe Leu Leu Ile Gln Ala Trp Pro Glu Asn Arg Thr Asp
405 410 415
Leu His Ala Phe Glu Asn Leu Glu Ile Ile Arg Gly Arg Thr Lys Gln
420 425 430
His Gly Gln Phe Ser Leu Ala Val Val Ser Leu Asn Ile Thr Ser Leu
435 440 445
Gly Leu Arg Ser Leu Lys Glu Ile Ser Asp Gly Asp Val Ile Ile Ser
450 455 460
Gly Asn Lys Asn Leu Cys Tyr Ala Asn Thr Ile Asn Trp Lys Lys Leu
465 470 475 480
Phe Gly Thr Ser Ser Gln Lys Thr Lys Ile Ile Ser Asn Arg Gly Glu
485 490 495
Asn Ser Cys Lys Ala Thr Gly Gln Val Cys His Ala Leu Cys Ser Pro
500 505 510
Glu Gly Cys Trp Gly Pro Glu Pro Arg Asp Cys Val Ser Cys Gln Asn
515 520 525
Val Ser Arg Gly Arg Glu Cys Val Asp Lys Cys Asn Ile Leu Glu Gly
530 535 540
Glu Pro Arg Glu Phe Val Glu Asn Ser Glu Cys Ile Gln Cys His Pro
545 550 555 560
Glu Cys Leu Pro Gln Val Met Asn Ile Thr Cys Thr Gly Arg Gly Pro
565 570 575
Asp Asn Cys Ile Gln Cys Ala His Tyr Ile Asp Gly Pro His Cys Val
580 585 590
Lys Thr Cys Pro Ala Gly Val Met Gly Glu Asn Asn Thr Leu Val Trp
595 600 605
Lys Tyr Ala Asp Ala Gly His Val Cys His Leu Cys His Pro Asn Cys
610 615 620
Thr Tyr Gly Cys Thr Gly Pro Gly Leu Glu Gly Cys Ala Arg
625 630 635
<210> 13
<211> 3630
<212> DNA
<213> Homo sapiens
<400> 13
atgcgaccct ccgggacggc cggggcagcg ctcctggcgc tgctggctgc gctctgcccg
60
gcgagtcggg ctctggagga aaagaaagtt tgccaaggca cgagtaacaa gctcacgcag
120
ttgggcactt ttgaagatca ttttctcagc ctccagagga tgttcaataa ctgtgaggtg
180
gtccttggga atttggaaat tacctatgtg cagaggaatt atgatctttc cttcttaaag
240
accatccagg aggtggctgg ttatgtcctc attgccctca acacagtgga gcgaattcct
300
ttggaaaacc tgcagatcat cagaggaaat atgtactacg aaaattccta tgccttagca
360
gtcttatcta actatgatgc aaataaaacc ggactgaagg agctgcccat gagaaattta
420
caggaaatcc tgcatggcgc cgtgcggttc agcaacaacc ctgccctgtg caacgtggag
480
agcatccagt ggcgggacat agtcagcagt gactttctca gcaacatgtc gatggacttc
540
cagaaccacc tgggcagctg ccaaaagtgt gatccaagct gtcccaatgg gagctgctgg
600
ggtgcaggag aggagaactg ccagaaactg accaaaatca tctgtgccca gcagtgctcc
660
gggcgctgcc gtggcaagtc ccccagtgac tgctgccaca accagtgtgc tgcaggctgc
720
acaggccccc gggagagcga ctgcctggtc tgccgcaaat tccgagacga agccacgtgc
780
aaggacacct gccccccact catgctctac aaccccacca cgtaccagat ggatgtgaac
840
cccgagggca aatacagctt tggtgccacc tgcgtgaaga agtgtccccg taattatgtg
900
gtgacagatc acggctcgtg cgtccgagcc tgtggggccg acagctatga gatggaggaa
960
gacggcgtcc gcaagtgtaa gaagtgcgaa gggccttgcc gcaaagtgtg taacggaata
1020
ggtattggtg aatttaaaga ctcactctcc ataaatgcta cgaatattaa acacttcaaa
1080
aactgcacct ccatcagtgg cgatctccac atcctgccgg tggcatttag gggtgactcc
1140
ttcacacata ctcctcctct ggatccacag gaactggata ttctgaaaac cgtaaaggaa
1200
atcacagggt ttttgctgat tcaggcttgg cctgaaaaca ggacggacct ccatgccttt
1260
gagaacctag aaatcatacg cggcaggacc aagcaacatg gtcagttttc tcttgcagtc
1320
gtcagcctga acataacatc cttgggatta cgctccctca aggagataag tgatggagat
1380
gtgataattt caggaaacaa aaatttgtgc tatgcaaata caataaactg gaaaaaactg
1440
tttgggacct ccggtcagaa aaccaaaatt ataagcaaca gaggtgaaaa cagctgcaag
1500
gccacaggcc aggtctgcca tgccttgtgc tcccccgagg gctgctgggg cccggagccc
1560
agggactgcg tctcttgccg gaatgtcagc cgaggcaggg aatgcgtgga caagtgcaac
1620
cttctggagg gtgagccaag ggagtttgtg gagaactctg agtgcataca gtgccaccca
1680
gagtgcctgc ctcaggccat gaacatcacc tgcacaggac ggggaccaga caactgtatc
1740
cagtgtgccc actacattga cggcccccac tgcgtcaaga cctgcccggc aggagtcatg
1800
ggagaaaaca acaccctggt ctggaagtac gcagacgccg gccatgtgtg ccacctgtgc
1860
catccaaact gcacctacgg atgcactggg ccaggtcttg aaggctgtcc aacgaatggg
1920
cctaagatcc cgtccatcgc cactgggatg gtgggggccc tcctcttgct gctggtggtg
1980
gccctgggga tcggcctctt catgcgaagg cgccacatcg ttcggaagcg cacgctgcgg
2040
aggctgctgc aggagaggga gcttgtggag cctcttacac ccagtggaga agctcccaac
2100
caagctctct tgaggatctt gaaggaaact gaattcaaaa agatcaaagt gctgggctcc
2160
ggtgcgttcg gcacggtgta taagggactc tggatcccag aaggtgagaa agttaaaatt
2220
cccgtcgcta tcaaggaatt aagagaagca acatctccga aagccaacaa ggaaatcctc
2280
gatgaagcct acgtgatggc cagcgtggac aacccccacg tgtgccgcct gctgggcatc
2340
tgcctcacct ccaccgtgca gctcatcacg cagctcatgc ccttcggctg cctcctggac
2400
tatgtccggg aacacaaaga caatattggc tcccagtacc tgctcaactg gtgtgtgcag
2460
atcgcaaagg gcatgaacta cttggaggac cgtcgcttgg tgcaccgcga cctggcagcc
2520
aggaacgtac tggtgaaaac accgcagcat gtcaagatca cagattttgg gctggccaaa
2580
ctgctgggtg cggaagagaa agaataccat gcagaaggag gcaaagtgcc tatcaagtgg
2640
atggcattgg aatcaatttt acacagaatc tatacccacc agagtgatgt ctggagctac
2700
ggggtgactg tttgggagtt gatgaccttt ggatccaagc catatgacgg aatccctgcc
2760
agcgagatct cctccatcct ggagaaagga gaacgcctcc ctcagccacc catatgtacc
2820
atcgatgtct acatgatcat ggtcaagtgc tggatgatag acgcagatag tcgcccaaag
2880
ttccgtgagt tgatcatcga attctccaaa atggcccgag acccccagcg ctaccttgtc
2940
attcaggggg atgaaagaat gcatttgcca agtcctacag actccaactt ctaccgtgcc
3000
ctgatggatg aagaagacat ggacgacgtg gtggatgccg acgagtacct catcccacag
3060
cagggcttct tcagcagccc ctccacgtca cggactcccc tcctgagctc tctgagtgca
3120
accagcaaca attccaccgt ggcttgcatt gatagaaatg ggctgcaaag ctgtcccatc
3180
aaggaagaca gcttcttgca gcgatacagc tcagacccca caggcgcctt gactgaggac
3240
agcatagacg acaccttcct cccagtgcct gaatacataa accagtccgt tcccaaaagg
3300
cccgctggct ctgtgcagaa tcctgtctat cacaatcagc ctctgaaccc cgcgcccagc
3360
agagacccac actaccagga cccccacagc actgcagtgg gcaaccccga gtatctcaac
3420
actgtccagc ccacctgtgt caacagcaca ttcgacagcc ctgcccactg ggcccagaaa
3480
ggcagccacc aaattagcct ggacaaccct gactaccagc aggacttctt tcccaaggaa
3540
gccaagccaa atggcatctt taagggctcc acagctgaaa atgcagaata cctaagggtc
3600
gcgccacaaa gcagtgaatt tattggagca
3630
<210> 14
<211> 1210
<212> PRT
<213> Homo sapiens
<400> 14
Met Arg Pro Ser Gly Thr Ala Gly Ala Ala Leu Leu Ala Leu Leu Ala
1 5 10 15
Ala Leu Cys Pro Ala Ser Arg Ala Leu Glu Glu Lys Lys Val Cys Gln
20 25 30
Gly Thr Ser Asn Lys Leu Thr Gln Leu Gly Thr Phe Glu Asp His Phe
35 40 45
Leu Ser Leu Gln Arg Met Phe Asn Asn Cys Glu Val Val Leu Gly Asn
50 55 60
Leu Glu Ile Thr Tyr Val Gln Arg Asn Tyr Asp Leu Ser Phe Leu Lys
65 70 75 80
Thr Ile Gln Glu Val Ala Gly Tyr Val Leu Ile Ala Leu Asn Thr Val
85 90 95
Glu Arg Ile Pro Leu Glu Asn Leu Gln Ile Ile Arg Gly Asn Met Tyr
100 105 110
Tyr Glu Asn Ser Tyr Ala Leu Ala Val Leu Ser Asn Tyr Asp Ala Asn
115 120 125
Lys Thr Gly Leu Lys Glu Leu Pro Met Arg Asn Leu Gln Glu Ile Leu
130 135 140
His Gly Ala Val Arg Phe Ser Asn Asn Pro Ala Leu Cys Asn Val Glu
145 150 155 160
Ser Ile Gln Trp Arg Asp Ile Val Ser Ser Asp Phe Leu Ser Asn Met
165 170 175
Ser Met Asp Phe Gln Asn His Leu Gly Ser Cys Gln Lys Cys Asp Pro
180 185 190
Ser Cys Pro Asn Gly Ser Cys Trp Gly Ala Gly Glu Glu Asn Cys Gln
195 200 205
Lys Leu Thr Lys Ile Ile Cys Ala Gln Gln Cys Ser Gly Arg Cys Arg
210 215 220
Gly Lys Ser Pro Ser Asp Cys Cys His Asn Gln Cys Ala Ala Gly Cys
225 230 235 240
Thr Gly Pro Arg Glu Ser Asp Cys Leu Val Cys Arg Lys Phe Arg Asp
245 250 255
Glu Ala Thr Cys Lys Asp Thr Cys Pro Pro Leu Met Leu Tyr Asn Pro
260 265 270
Thr Thr Tyr Gln Met Asp Val Asn Pro Glu Gly Lys Tyr Ser Phe Gly
275 280 285
Ala Thr Cys Val Lys Lys Cys Pro Arg Asn Tyr Val Val Thr Asp His
290 295 300
Gly Ser Cys Val Arg Ala Cys Gly Ala Asp Ser Tyr Glu Met Glu Glu
305 310 315 320
Asp Gly Val Arg Lys Cys Lys Lys Cys Glu Gly Pro Cys Arg Lys Val
325 330 335
Cys Asn Gly Ile Gly Ile Gly Glu Phe Lys Asp Ser Leu Ser Ile Asn
340 345 350
Ala Thr Asn Ile Lys His Phe Lys Asn Cys Thr Ser Ile Ser Gly Asp
355 360 365
Leu His Ile Leu Pro Val Ala Phe Arg Gly Asp Ser Phe Thr His Thr
370 375 380
Pro Pro Leu Asp Pro Gln Glu Leu Asp Ile Leu Lys Thr Val Lys Glu
385 390 395 400
Ile Thr Gly Phe Leu Leu Ile Gln Ala Trp Pro Glu Asn Arg Thr Asp
405 410 415
Leu His Ala Phe Glu Asn Leu Glu Ile Ile Arg Gly Arg Thr Lys Gln
420 425 430
His Gly Gln Phe Ser Leu Ala Val Val Ser Leu Asn Ile Thr Ser Leu
435 440 445
Gly Leu Arg Ser Leu Lys Glu Ile Ser Asp Gly Asp Val Ile Ile Ser
450 455 460
Gly Asn Lys Asn Leu Cys Tyr Ala Asn Thr Ile Asn Trp Lys Lys Leu
465 470 475 480
Phe Gly Thr Ser Gly Gln Lys Thr Lys Ile Ile Ser Asn Arg Gly Glu
485 490 495
Asn Ser Cys Lys Ala Thr Gly Gln Val Cys His Ala Leu Cys Ser Pro
500 505 510
Glu Gly Cys Trp Gly Pro Glu Pro Arg Asp Cys Val Ser Cys Arg Asn
515 520 525
Val Ser Arg Gly Arg Glu Cys Val Asp Lys Cys Asn Leu Leu Glu Gly
530 535 540
Glu Pro Arg Glu Phe Val Glu Asn Ser Glu Cys Ile Gln Cys His Pro
545 550 555 560
Glu Cys Leu Pro Gln Ala Met Asn Ile Thr Cys Thr Gly Arg Gly Pro
565 570 575
Asp Asn Cys Ile Gln Cys Ala His Tyr Ile Asp Gly Pro His Cys Val
580 585 590
Lys Thr Cys Pro Ala Gly Val Met Gly Glu Asn Asn Thr Leu Val Trp
595 600 605
Lys Tyr Ala Asp Ala Gly His Val Cys His Leu Cys His Pro Asn Cys
610 615 620
Thr Tyr Gly Cys Thr Gly Pro Gly Leu Glu Gly Cys Pro Thr Asn Gly
625 630 635 640
Pro Lys Ile Pro Ser Ile Ala Thr Gly Met Val Gly Ala Leu Leu Leu
645 650 655
Leu Leu Val Val Ala Leu Gly Ile Gly Leu Phe Met Arg Arg Arg His
660 665 670
Ile Val Arg Lys Arg Thr Leu Arg Arg Leu Leu Gln Glu Arg Glu Leu
675 680 685
Val Glu Pro Leu Thr Pro Ser Gly Glu Ala Pro Asn Gln Ala Leu Leu
690 695 700
Arg Ile Leu Lys Glu Thr Glu Phe Lys Lys Ile Lys Val Leu Gly Ser
705 710 715 720
Gly Ala Phe Gly Thr Val Tyr Lys Gly Leu Trp Ile Pro Glu Gly Glu
725 730 735
Lys Val Lys Ile Pro Val Ala Ile Lys Glu Leu Arg Glu Ala Thr Ser
740 745 750
Pro Lys Ala Asn Lys Glu Ile Leu Asp Glu Ala Tyr Val Met Ala Ser
755 760 765
Val Asp Asn Pro His Val Cys Arg Leu Leu Gly Ile Cys Leu Thr Ser
770 775 780
Thr Val Gln Leu Ile Thr Gln Leu Met Pro Phe Gly Cys Leu Leu Asp
785 790 795 800
Tyr Val Arg Glu His Lys Asp Asn Ile Gly Ser Gln Tyr Leu Leu Asn
805 810 815
Trp Cys Val Gln Ile Ala Lys Gly Met Asn Tyr Leu Glu Asp Arg Arg
820 825 830
Leu Val His Arg Asp Leu Ala Ala Arg Asn Val Leu Val Lys Thr Pro
835 840 845
Gln His Val Lys Ile Thr Asp Phe Gly Leu Ala Lys Leu Leu Gly Ala
850 855 860
Glu Glu Lys Glu Tyr His Ala Glu Gly Gly Lys Val Pro Ile Lys Trp
865 870 875 880
Met Ala Leu Glu Ser Ile Leu His Arg Ile Tyr Thr His Gln Ser Asp
885 890 895
Val Trp Ser Tyr Gly Val Thr Val Trp Glu Leu Met Thr Phe Gly Ser
900 905 910
Lys Pro Tyr Asp Gly Ile Pro Ala Ser Glu Ile Ser Ser Ile Leu Glu
915 920 925
Lys Gly Glu Arg Leu Pro Gln Pro Pro Ile Cys Thr Ile Asp Val Tyr
930 935 940
Met Ile Met Val Lys Cys Trp Met Ile Asp Ala Asp Ser Arg Pro Lys
945 950 955 960
Phe Arg Glu Leu Ile Ile Glu Phe Ser Lys Met Ala Arg Asp Pro Gln
965 970 975
Arg Tyr Leu Val Ile Gln Gly Asp Glu Arg Met His Leu Pro Ser Pro
980 985 990
Thr Asp Ser Asn Phe Tyr Arg Ala Leu Met Asp Glu Glu Asp Met Asp
995 1000 1005
Asp Val Val Asp Ala Asp Glu Tyr Leu Ile Pro Gln Gln Gly Phe
1010 1015 1020
Phe Ser Ser Pro Ser Thr Ser Arg Thr Pro Leu Leu Ser Ser Leu
1025 1030 1035
Ser Ala Thr Ser Asn Asn Ser Thr Val Ala Cys Ile Asp Arg Asn
1040 1045 1050
Gly Leu Gln Ser Cys Pro Ile Lys Glu Asp Ser Phe Leu Gln Arg
1055 1060 1065
Tyr Ser Ser Asp Pro Thr Gly Ala Leu Thr Glu Asp Ser Ile Asp
1070 1075 1080
Asp Thr Phe Leu Pro Val Pro Glu Tyr Ile Asn Gln Ser Val Pro
1085 1090 1095
Lys Arg Pro Ala Gly Ser Val Gln Asn Pro Val Tyr His Asn Gln
1100 1105 1110
Pro Leu Asn Pro Ala Pro Ser Arg Asp Pro His Tyr Gln Asp Pro
1115 1120 1125
His Ser Thr Ala Val Gly Asn Pro Glu Tyr Leu Asn Thr Val Gln
1130 1135 1140
Pro Thr Cys Val Asn Ser Thr Phe Asp Ser Pro Ala His Trp Ala
1145 1150 1155
Gln Lys Gly Ser His Gln Ile Ser Leu Asp Asn Pro Asp Tyr Gln
1160 1165 1170
Gln Asp Phe Phe Pro Lys Glu Ala Lys Pro Asn Gly Ile Phe Lys
1175 1180 1185
Gly Ser Thr Ala Glu Asn Ala Glu Tyr Leu Arg Val Ala Pro Gln
1190 1195 1200
Ser Ser Glu Phe Ile Gly Ala
1205 1210
<210> 15
<211> 327
<212> DNA
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the base sequence of
A27
VL
<400> 15
gaaatagtgt tgacgcagtc tccaggcacc ctgtctttgt ctccagggga aagagccacc
60
ctctcctgca gggccagtca gagtgttagc agcagctact tagcctggta ccagcagaaa
120
cctggccagg ctcccaggct cctcatctat ggtgcatcca gcagggccac tggcatccca
180
gacaggttca gtggcagtgg gtctgggaca gacttcactc tcaccatcag cagactggag
240
cctgaagatt ttgcagtgta ttactgtcag cagtatggta gctcacctcc gtggacgttc
300
ggccaaggga ccaaggtgga aatcaaa
327
<210> 16
<211> 109
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of A27 VL
<400> 16
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Ser Ser Pro
85 90 95
Pro Trp Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 17
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of A27 LCDR1
<400> 17
Arg Ala Ser Gln Ser Val Ser Ser Ser Tyr Leu Ala
1 5 10
<210> 18
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of A27 LCDR2
<400> 18
Gly Ala Ser Ser Arg Ala Thr
1 5
<210> 19
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of A27 LCDR3
<400> 19
Gln Gln Tyr Gly Ser Ser Pro Pro Trp Thr
1 5 10
<210> 20
<211> 366
<212> DNA
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the base sequence of
PSM4072 VH
<400> 20
gaggtgcagc tggtggagac tgggggaggc ttggtgcaac ctggggggtc cctgagactc
60
tcctgtacag cctctggatt cacctttgaa agtcatgcca tgtactgggt ccggcaagct
120
ccagggaagg ggctggagtg ggtctcgggt attagtaatg gaggtagtag cacagagtac
180
gcagactccg tgaggggccg gttcaccatt tccagagaca attccaagaa tacggtgtat
240
ctgcaaatga acagcctgag agccgaggac acggccgtat attactgtgc gagaccaaga
300
tctccatatc tcttctacga tgctgctggt gtctggggcc aagggaccct ggtcaccgtc
360
tcctca
366
<210> 21
<211> 122
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of PSM4072 VH
<400> 21
Glu Val Gln Leu Val Glu Thr Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Thr Phe Glu Ser His
20 25 30
Ala Met Tyr Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Asn Gly Gly Ser Ser Thr Glu Tyr Ala Asp Ser Val
50 55 60
Arg Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Val Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Pro Arg Ser Pro Tyr Leu Phe Tyr Asp Ala Ala Gly Val Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 22
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of PSM4072 HCDR1
<400> 22
Ser His Ala Met Tyr
1 5
<210> 23
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of PSM4072 HCDR2
<400> 23
Gly Ile Ser Asn Gly Gly Ser Ser Thr Glu Tyr Ala Asp Ser Val Arg
1 5 10 15
Gly
<210> 24
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of PSM4072 HCDR3
<400> 24
Pro Arg Ser Pro Tyr Leu Phe Tyr Asp Ala Ala Gly Val
1 5 10
<210> 25
<211> 366
<212> DNA
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the base sequence of
R327
VH
<400> 25
gaggtgcagc tggtggagac cgggggaggc ttggtacagc ctggggggtc cctgagactc
60
tcctgtgcag cctctggatt cagctttgac aagtatacca tgaactgggt ccgccaggct
120
ccagggaagg ggctggagtg ggtctcagtt atttctaatg ggggagtttc tacagactac
180
gcagactccg ttaagggccg gttcaccgtc tccagagaca attccaagaa cacactgtac
240
ctgcaaatga acagcctgag agccgaggat atggccacat attactgtgc gcgtgcgagc
300
cagccgtggc tctataggac cggtgcggat gtgtggggcc aggggacaat ggtcaccgtc
360
tcttca
366
<210> 26
<211> 122
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of R327 VH
<400> 26
Glu Val Gln Leu Val Glu Thr Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser Phe Asp Lys Tyr
20 25 30
Thr Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Val Ile Ser Asn Gly Gly Val Ser Thr Asp Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Val Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Met Ala Thr Tyr Tyr Cys
85 90 95
Ala Arg Ala Ser Gln Pro Trp Leu Tyr Arg Thr Gly Ala Asp Val Trp
100 105 110
Gly Gln Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 27
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of R327 HCDR1
<400> 27
Lys Tyr Thr Met Asn
1 5
<210> 28
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of R327 HCDR2
<400> 28
Val Ile Ser Asn Gly Gly Val Ser Thr Asp Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 29
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of R327 HCDR3
<400> 29
Ala Ser Gln Pro Trp Leu Tyr Arg Thr Gly Ala Asp Val
1 5 10
<210> 30
<211> 366
<212> DNA
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the base sequence of
TfR1071 VH
<400> 30
tacgtgcagc tggtggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc
60
tcctgtgcag cctctggatt cagctttgac aagtatacca tgaactgggt ccgccaggct
120
ccagggaagg ggctggagtg ggtctcagtt atttctaatg ggggagtttc tacagactac
180
gcagactccg ttaagggccg gttcaccgtc tccagagaca attccaagaa cacactgtac
240
ctgcaaatga acagcctgag agccgaggat atggccacat attactgtgc gcgtgcgagc
300
cagccgtggc tctataggac cggtgcggat gtgtggggcc aggggaccct ggtcaccgtc
360
tcctca
366
<210> 31
<211> 122
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of TfR1071 VH
<400> 31
Tyr Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser Phe Asp Lys Tyr
20 25 30
Thr Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Val Ile Ser Asn Gly Gly Val Ser Thr Asp Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Val Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Met Ala Thr Tyr Tyr Cys
85 90 95
Ala Arg Ala Ser Gln Pro Trp Leu Tyr Arg Thr Gly Ala Asp Val Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 32
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of TfR1071 HCDR1
<400> 32
Lys Tyr Thr Met Asn
1 5
<210> 33
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of TfR1071 HCDR2
<400> 33
Val Ile Ser Asn Gly Gly Val Ser Thr Asp Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 34
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of TfR1071 HCDR3
<400> 34
Ala Ser Gln Pro Trp Leu Tyr Arg Thr Gly Ala Asp Val
1 5 10
<210> 35
<211> 366
<212> DNA
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the base sequence of
TfR4016 VH
<400> 35
tacgtgcagc tggtggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc
60
tcctgtgcag cctctggatt cagctttgac aagtatacca tgaactgggt ccgccaggct
120
ccagggaagg ggctggagtg ggtctcagtt atttctaatg ggggagtttc tacagactac
180
gcagactccg ttaagggccg gttcaccgtc tccagagaca attccaagaa cacactgtac
240
ctgcaaatga acagcctgag agccgaggat atggccacat attactgtgc gcgtgcgagc
300
cagccgtggc tctataggac cggtgcggat gtgtggggcc aggggaccac ggtcaccgtc
360
tcctca
366
<210> 36
<211> 122
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of TfR4016 VH
<400> 36
Tyr Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser Phe Asp Lys Tyr
20 25 30
Thr Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Val Ile Ser Asn Gly Gly Val Ser Thr Asp Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Val Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Met Ala Thr Tyr Tyr Cys
85 90 95
Ala Arg Ala Ser Gln Pro Trp Leu Tyr Arg Thr Gly Ala Asp Val Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 37
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of TfR4016 HCDR1
<400> 37
Lys Tyr Thr Met Asn
1 5
<210> 38
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of TfR4016 HCDR2
<400> 38
Val Ile Ser Asn Gly Gly Val Ser Thr Asp Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 39
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of TfR4016 HCDR3
<400> 39
Ala Ser Gln Pro Trp Leu Tyr Arg Thr Gly Ala Asp Val
1 5 10
<210> 40
<211> 366
<212> DNA
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the base sequence of
cyno186 VH
<400> 40
caggtgcagc tggtggagtc tgggggagaa ttcgtacagc ctggggggtc cctgagactc
60
tcctgtgcag cctctggatt cccctttaaa ggctatgcca tgaattgggt ccgccaggct
120
ccagggaagg gattggagtg ggtctcaaga ataagtaatg gtggtagcta catagactac
180
gcagactccg taaagggccg gttcaccatc tccagagaca attccaagaa cacgctgtat
240
ttgcaaatag acagcctgag aaccgaggac acggccgtat attactgtgc gaaagcgaag
300
gacgcctata ggtggaacaa cgctatagac gaatggggcc aagggaccct ggtcaccgtc
360
tcctca
366
<210> 41
<211> 122
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of cyno186 VH
<400> 41
Gln Val Gln Leu Val Glu Ser Gly Gly Glu Phe Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Pro Phe Lys Gly Tyr
20 25 30
Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Arg Ile Ser Asn Gly Gly Ser Tyr Ile Asp Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Ile Asp Ser Leu Arg Thr Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Ala Lys Asp Ala Tyr Arg Trp Asn Asn Ala Ile Asp Glu Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 42
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of cyno186 HCDR1
<400> 42
Gly Tyr Ala Met Asn
1 5
<210> 43
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of cyno186 HCDR2
<400> 43
Arg Ile Ser Asn Gly Gly Ser Tyr Ile Asp Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 44
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of cyno186 HCDR3
<400> 44
Ala Lys Asp Ala Tyr Arg Trp Asn Asn Ala Ile Asp Glu
1 5 10
<210> 45
<211> 366
<212> DNA
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the base sequence of
cyno292 VH
<400> 45
caggtgcagc tggtgcaatc tgggggaggc ctggtcaagc ctggggggtc cctgagaatc
60
tcctgtacaa cctctggatt cccctttgat ggctatacca tgacctgggt ccgccaggct
120
ccagggaagg ggctggagtg ggtctcaagt attagtaatg gtggtagcac cattttcgtc
180
tcagactcag tgagaggccg attcaccatc tccagagaca acgccaagaa ttcactgtat
240
ctccaaatga acaacgtggg agccgaggac acggctgtgt attactgtgc gagagctcga
300
gatgcgtatg gctggacgct tccttctgac atctggggcc aagggacaat ggtcaccgtc
360
tcttca
366
<210> 46
<211> 122
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of cyno292 VH
<400> 46
Gln Val Gln Leu Val Gln Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Ile Ser Cys Thr Thr Ser Gly Phe Pro Phe Asp Gly Tyr
20 25 30
Thr Met Thr Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ser Ile Ser Asn Gly Gly Ser Thr Ile Phe Val Ser Asp Ser Val
50 55 60
Arg Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Asn Val Gly Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ala Arg Asp Ala Tyr Gly Trp Thr Leu Pro Ser Asp Ile Trp
100 105 110
Gly Gln Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 47
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of cyno292 HCDR1
<400> 47
Gly Tyr Thr Met Thr
1 5
<210> 48
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of cyno292 HCDR2
<400> 48
Ser Ile Ser Asn Gly Gly Ser Thr Ile Phe Val Ser Asp Ser Val Arg
1 5 10 15
Gly
<210> 49
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of cyno292 HCDR3
<400> 49
Ala Arg Asp Ala Tyr Gly Trp Thr Leu Pro Ser Asp Ile
1 5 10
<210> 50
<211> 122
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of TfR1007 VH
<400> 50
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Ser Leu Ser Cys Ala Ala Ser Gly Phe Ser Phe Lys Gly Tyr
20 25 30
Ser Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ser Leu Ser Asn Gly Gly Gly Ser Ser Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Thr Leu Gly Ala Tyr Tyr Ile Lys Ser Ala Met Asp Val Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 51
<211> 122
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of cyno163 VH
<400> 51
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Thr Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Asn Gly Gly Ser Ser Thr Glu Tyr Ala Asp Ser Val
50 55 60
Arg Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Val Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Pro Arg Ser Pro Tyr Leu Phe Tyr Asp Ala Ala Gly Val Trp
100 105 110
Gly Gln Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 52
<211> 123
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of 22-30 VH
<400> 52
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Tyr
20 25 30
Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Asn Tyr Asn Pro Ser Leu Lys
50 55 60
Ser Arg Val Thr Ile Ser Leu Asp Thr Ser Lys Asn Gln Phe Ser Leu
65 70 75 80
Arg Leu Ser Ser Val Thr Thr Ala Asp Thr Ala Leu Tyr Tyr Cys Thr
85 90 95
Arg Glu Ala Gly Tyr Tyr Asp Ser Ser Gly Tyr Tyr Gly Phe Asp Tyr
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 53
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of 22-30 VL
<400> 53
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Glu Lys Ala Pro Lys Ser Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Phe Cys Gln Gln Tyr Tyr Asn Tyr Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 54
<211> 114
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of T14 VH
<400> 54
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Val Val Ser Gly Gly Thr Phe Ser Asn Phe
20 25 30
Asn Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Tyr Ile Ser Gly Ser Ser Ser Thr Ile Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Asp Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Ser Ser Ser Trp Tyr Tyr Trp Gly Gln Gly Thr Leu Val Thr Val
100 105 110
Ser Ser
<210> 55
<211> 123
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of TfR434 VH
<400> 55
Gln Val Gln Leu Val Gln Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Pro Phe Lys Ser Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Ser Tyr Asp Gly Ser Ser Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Gly Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Ser Asn Phe Trp Ser Gly Tyr Tyr Ser Pro Val Asp Val
100 105 110
Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 56
<211> 123
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of TfR435 VH
<400> 56
Gln Val Gln Leu Val Gln Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Pro Phe Lys Ser Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Ser Phe Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Gly Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Ser Asn Phe Trp Ser Gly Tyr Tyr Ser Pro Val Asp Val
100 105 110
Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 57
<211> 111
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of TfR434,435 VL
<400> 57
Asn Phe Met Leu Thr Gln Pro His Ser Val Ser Glu Ser Pro Gly Lys
1 5 10 15
Thr Val Thr Ile Ser Cys Thr Arg Ser Ser Gly Ser Ile Ala Ser Asn
20 25 30
Ser Val Gln Trp Tyr Gln Gln Arg Pro Gly Ser Ala Pro Ile Thr Val
35 40 45
Ile Tyr Glu Asp Thr Gln Arg Pro Ser Gly Val Pro Asp Arg Phe Ser
50 55 60
Gly Ser Ile Asp Ser Ser Ser Asn Ser Ala Ser Leu Thr Ile Ser Gly
65 70 75 80
Leu Gln Thr Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Ser
85 90 95
Ala Tyr His Trp Val Phe Gly Gly Gly Thr Lys Leu Ala Val Leu
100 105 110
<210> 58
<211> 366
<212> DNA
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the base sequence of
E08
VH
<400> 58
gaggtgcagc tggtggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc
60
tcctgtgcag cctctggatt cagctttaac aacaatgcca tgaactgggt ccgtcaggct
120
ccagggaagg ggctggagtg ggtctcaggt attcgtgccg atggcggtac gacatactac
180
gcagactccg tgaagggccg gttcaccatc tccagagaca attccaagaa cacactgtat
240
ctgcaaatga acagcctgag agccgaggac acggccgtat atttctgtgg aaagacgcct
300
gactgggaaa tcttctacta cgctatggac gcctggggcc aagggaccac ggtcaccgtc
360
tcctca
366
<210> 59
<211> 122
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of E08 VH
<400> 59
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser Phe Asn Asn Asn
20 25 30
Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Arg Ala Asp Gly Gly Thr Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Phe Cys
85 90 95
Gly Lys Thr Pro Asp Trp Glu Ile Phe Tyr Tyr Ala Met Asp Ala Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 60
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of E08 HCDR1
<400> 60
Asn Asn Ala Met Asn
1 5
<210> 61
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of E08 HCDR2
<400> 61
Gly Ile Arg Ala Asp Gly Gly Thr Thr Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 62
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of E08 HCDR3
<400> 62
Thr Pro Asp Trp Glu Ile Phe Tyr Tyr Ala Met Asp Ala
1 5 10
<210> 63
<211> 363
<212> DNA
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the base sequence of
E12
VH
<400> 63
caggtgcagc tggtggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc
60
tcctgtgcag cctctagatt cacctttagc gcctatgcca tgggctgggt gcgccaggct
120
ccagggaagg ggctggagtg ggtctcaatt attgatagtg gtggtgtgta cacatactac
180
gcagactcca tgaagggccg cttcaccatc tccagagaca attccaagaa cacgctgtat
240
ctgcaaatga acagcctgag agccgaggac acggccgtat attactgtgc gagagatcat
300
tatggtgttg tttggggact tggagattac tggggccagg gaaccctggt cactgtctcc
360
tca
363
<210> 64
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of E12 VH
<400> 64
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Arg Phe Thr Phe Ser Ala Tyr
20 25 30
Ala Met Gly Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ile Ile Asp Ser Gly Gly Val Tyr Thr Tyr Tyr Ala Asp Ser Met
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp His Tyr Gly Val Val Trp Gly Leu Gly Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 65
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of E12 HCDR1
<400> 65
Ala Tyr Ala Met Gly
1 5
<210> 66
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of E12 HCDR2
<400> 66
Ile Ile Asp Ser Gly Gly Val Tyr Thr Tyr Tyr Ala Asp Ser Met Lys
1 5 10 15
Gly
<210> 67
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of E12 HCDR3
<400> 67
Asp His Tyr Gly Val Val Trp Gly Leu Gly Asp Tyr
1 5 10
<210> 68
<211> 354
<212> DNA
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the base sequence of
E17
VH
<400> 68
caggtgcagc tacagcagtg gggcgcagga ctgttgaagc cttcggagac tctgtccctc
60
acctgcgcac tgtacagtgg gtccttcagt gctcaggact ggagctggat ccgccagtcc
120
ccagagaagg ggctggagtg gattggggaa atctggcaag ggggaaaaac caactacaac
180
ccgtccctca agagtcgagt tagtatatca agagacaact ccaagaacca gttgtccctg
240
cagctgagct ctgtgaccgc cgcggacacg gctgtgtatt actgtgcgag ggtggactgg
300
tcttatcgtg tttttgaaat ctggggccaa gggacaatgg tcaccgtctc ttca
354
<210> 69
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of E17 VH
<400> 69
Gln Val Gln Leu Gln Gln Trp Gly Ala Gly Leu Leu Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Ala Leu Tyr Ser Gly Ser Phe Ser Ala Gln
20 25 30
Asp Trp Ser Trp Ile Arg Gln Ser Pro Glu Lys Gly Leu Glu Trp Ile
35 40 45
Gly Glu Ile Trp Gln Gly Gly Lys Thr Asn Tyr Asn Pro Ser Leu Lys
50 55 60
Ser Arg Val Ser Ile Ser Arg Asp Asn Ser Lys Asn Gln Leu Ser Leu
65 70 75 80
Gln Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Val Asp Trp Ser Tyr Arg Val Phe Glu Ile Trp Gly Gln Gly Thr
100 105 110
Met Val Thr Val Ser Ser
115
<210> 70
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of E17 HCDR1
<400> 70
Ala Gln Asp Trp Ser
1 5
<210> 71
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of E17 HCDR2
<400> 71
Glu Ile Trp Gln Gly Gly Lys Thr Asn Tyr Asn Pro Ser Leu Lys Ser
1 5 10 15
<210> 72
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of E17 HCDR3
<400> 72
Val Asp Trp Ser Tyr Arg Val Phe Glu Ile
1 5 10
<210> 73
<211> 123
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of KME07 VH
<400> 73
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Phe
20 25 30
Ala Phe Gly Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Glu Val Arg Pro Gly Phe Gly Thr Glu Ile Leu Thr Gln Asn Phe
50 55 60
Gln Asp Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Thr Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Lys Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Leu Leu Arg Gly Gly Tyr Ile Glu Asn Pro Phe Glu Ile
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 74
<211> 123
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of KME09 VH
<400> 74
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Phe
20 25 30
Ala Trp Gly Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Glu Ile Arg Pro Gly Phe Gly Thr Glu Ile Tyr Ala Gln Asn Phe
50 55 60
Gln Asp Arg Val Thr Phe Ile Thr Asp Glu Ala Thr Ser Thr Thr Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Lys Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Leu Leu Arg Ser Gly Tyr Ile Asp Asn Pro Cys Asp Ile
100 105 110
Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 75
<211> 125
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of KME11 VH
<400> 75
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Leu Ser Thr Tyr
20 25 30
Gly Ile Thr Trp Val Arg Gln Ala Pro Gly Gln Gly Phe Ala Trp Met
35 40 45
Gly Glu Ile Ile Pro Ser Leu Gly Ile Val Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Val Thr Tyr Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Gly Asp Glu Leu Ser Glu Gly His Ser Glu Tyr Tyr His Gly Met
100 105 110
Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120 125
<210> 76
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of Cetuximab VH
<400> 76
Gln Val Gln Leu Lys Gln Ser Gly Pro Gly Leu Val Gln Pro Ser Gln
1 5 10 15
Ser Leu Ser Ile Thr Cys Thr Val Ser Gly Phe Ser Leu Thr Asn Tyr
20 25 30
Gly Val His Trp Val Arg Gln Ser Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Val Ile Trp Ser Gly Gly Asn Thr Asp Tyr Asn Thr Pro Phe Thr
50 55 60
Ser Arg Leu Ser Ile Asn Lys Asp Asn Ser Lys Ser Gln Val Phe Phe
65 70 75 80
Lys Met Asn Ser Leu Gln Ser Asn Asp Thr Ala Ile Tyr Tyr Cys Ala
85 90 95
Arg Ala Leu Thr Tyr Tyr Asp Tyr Glu Phe Ala Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ala
115
<210> 77
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of Cetuximab VL
<400> 77
Asp Ile Leu Leu Thr Gln Ser Pro Val Ile Leu Ser Val Ser Pro Gly
1 5 10 15
Glu Arg Val Ser Phe Ser Cys Arg Ala Ser Gln Ser Ile Gly Thr Asn
20 25 30
Ile His Trp Tyr Gln Gln Arg Thr Asn Gly Ser Pro Arg Leu Leu Ile
35 40 45
Lys Tyr Ala Ser Glu Ser Ile Ser Gly Ile Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Ser Ile Asn Ser Val Glu Ser
65 70 75 80
Glu Asp Ile Ala Asp Tyr Tyr Cys Gln Gln Asn Asn Asn Trp Pro Thr
85 90 95
Thr Phe Gly Ala Gly Thr Lys Leu Glu Leu Lys
100 105
<210> 78
<211> 117
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of HN3 VH
<400> 78
Gln Val Gln Leu Val Gln Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Tyr Phe Asp Phe Asp Ser Tyr
20 25 30
Glu Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Ser Ile Tyr His Ser Gly Ser Thr Tyr Tyr Asn Pro Ser Leu Lys
50 55 60
Ser Arg Val Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu
65 70 75 80
Gln Met Asn Thr Leu Arg Ala Glu Asp Thr Ala Thr Tyr Tyr Cys Ala
85 90 95
Arg Val Asn Met Asp Arg Phe Asp Tyr Trp Gly Gln Gly Thr Leu Val
100 105 110
Thr Val Ser Ser Ser
115
<210> 79
<211> 2
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid of
sequence of linker
<400> 79
Glu Ser
1
<210> 80
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid of
sequence of linker
<400> 80
Glu Ser Lys Tyr Gly
1 5
<210> 81
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid of
sequence of linker
<400> 81
Glu Ser Lys Tyr Gly Pro Pro
1 5
<210> 82
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid of
sequence of linker
<400> 82
Gly Gly Gly Gly Ser
1 5
<210> 83
<211> 981
<212> DNA
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the base sequence of
IgG4PE R409K constant region
<400> 83
gctagcacca aggggccatc cgtcttcccc ctggcgccct gctccaggag cacctccgag
60
agcacagccg ccctgggctg cctggtcaag gactacttcc ccgaaccggt gacggtgtcg
120
tggaactcag gcgccctgac cagcggcgtg cacaccttcc cggctgtcct acagtcctca
180
ggactctact ccctcagcag cgtggtgacc gtgccctcca gcagcttggg cacgaagacc
240
tacacctgca acgtagatca caagcccagc aacaccaagg tggacaagag agttgagtcc
300
aaatatggtc ccccatgccc accatgccca gcacctgagt tcgagggggg accatcagtc
360
ttcctgttcc ccccaaaacc caaggacact ctcatgatct cccggacccc tgaggtcacg
420
tgcgtggtgg tggacgtgag ccaggaagac cccgaggtcc agttcaactg gtacgtggat
480
ggcgtggagg tgcataatgc caagacaaag ccgcgggagg agcagttcaa cagcacgtac
540
cgtgtggtca gcgtcctcac cgtcctgcac caggactggc tgaacggcaa ggagtacaag
600
tgcaaggtct ccaacaaagg cctcccgtcc tccatcgaga aaaccatctc caaagccaaa
660
gggcagcccc gagagccaca ggtgtacacc ctgcccccat cccaggagga gatgaccaag
720
aaccaggtca gcctgacctg cctggtcaaa ggcttctacc ccagcgacat cgccgtggag
780
tgggagagca atgggcagcc ggagaacaac tacaagacca cgcctcccgt gctggactcc
840
gacggctcct tcttcctcta cagcaagcta accgtggaca agagcaggtg gcaggagggg
900
aatgtcttct catgctccgt gatgcatgag gctctgcaca accactacac acagaagagc
960
ctctccctgt ctctgggtaa a
981
<210> 84
<211> 327
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of IgG4PE R409K constant region
<400> 84
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg
1 5 10 15
Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Lys Thr
65 70 75 80
Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Arg Val Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro Ala Pro
100 105 110
Glu Phe Glu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
115 120 125
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
130 135 140
Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp
145 150 155 160
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe
165 170 175
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
180 185 190
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu
195 200 205
Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
210 215 220
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys
225 230 235 240
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
245 250 255
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
260 265 270
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
275 280 285
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser
290 295 300
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
305 310 315 320
Leu Ser Leu Ser Leu Gly Lys
325
<210> 85
<211> 294
<212> DNA
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the base sequence of
modified codons of IgG4 CH1
<400> 85
gctagcacca aaggaccttc tgtatttcct cttgcgccat gctctcgctc tacgtcagaa
60
tcaactgccg ctctggggtg cctggttaaa gactacttcc cggagcctgt gacagtgagt
120
tggaactccg gcgccctgac atcaggagtg catacatttc ccgccgtgct tcagagcagc
180
ggactttata gcctcagcag tgtggtgacc gtgccatctt ccagcctggg gaccaagacc
240
tacacctgta acgtggacca caaacccagc aacaccaagg ttgataagag ggtc
294
<210> 86
<211> 327
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of IgG4PE constant region
<400> 86
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg
1 5 10 15
Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Lys Thr
65 70 75 80
Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Arg Val Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro Ala Pro
100 105 110
Glu Phe Glu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
115 120 125
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
130 135 140
Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp
145 150 155 160
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe
165 170 175
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
180 185 190
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu
195 200 205
Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
210 215 220
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys
225 230 235 240
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
245 250 255
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
260 265 270
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
275 280 285
Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser
290 295 300
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
305 310 315 320
Leu Ser Leu Ser Leu Gly Lys
325
<210> 87
<211> 763
<212> PRT
<213> Mus musculus
<400> 87
Met Met Asp Gln Ala Arg Ser Ala Phe Ser Asn Leu Phe Gly Gly Glu
1 5 10 15
Pro Leu Ser Tyr Thr Arg Phe Ser Leu Ala Arg Gln Val Asp Gly Asp
20 25 30
Asn Ser His Val Glu Met Lys Leu Ala Ala Asp Glu Glu Glu Asn Ala
35 40 45
Asp Asn Asn Met Lys Ala Ser Val Arg Lys Pro Lys Arg Phe Asn Gly
50 55 60
Arg Leu Cys Phe Ala Ala Ile Ala Leu Val Ile Phe Phe Leu Ile Gly
65 70 75 80
Phe Met Ser Gly Tyr Leu Gly Tyr Cys Lys Arg Val Glu Gln Lys Glu
85 90 95
Glu Cys Val Lys Leu Ala Glu Thr Glu Glu Thr Asp Lys Ser Glu Thr
100 105 110
Met Glu Thr Glu Asp Val Pro Thr Ser Ser Arg Leu Tyr Trp Ala Asp
115 120 125
Leu Lys Thr Leu Leu Ser Glu Lys Leu Asn Ser Ile Glu Phe Ala Asp
130 135 140
Thr Ile Lys Gln Leu Ser Gln Asn Thr Tyr Thr Pro Arg Glu Ala Gly
145 150 155 160
Ser Gln Lys Asp Glu Ser Leu Ala Tyr Tyr Ile Glu Asn Gln Phe His
165 170 175
Glu Phe Lys Phe Ser Lys Val Trp Arg Asp Glu His Tyr Val Lys Ile
180 185 190
Gln Val Lys Ser Ser Ile Gly Gln Asn Met Val Thr Ile Val Gln Ser
195 200 205
Asn Gly Asn Leu Asp Pro Val Glu Ser Pro Glu Gly Tyr Val Ala Phe
210 215 220
Ser Lys Pro Thr Glu Val Ser Gly Lys Leu Val His Ala Asn Phe Gly
225 230 235 240
Thr Lys Lys Asp Phe Glu Glu Leu Ser Tyr Ser Val Asn Gly Ser Leu
245 250 255
Val Ile Val Arg Ala Gly Glu Ile Thr Phe Ala Glu Lys Val Ala Asn
260 265 270
Ala Gln Ser Phe Asn Ala Ile Gly Val Leu Ile Tyr Met Asp Lys Asn
275 280 285
Lys Phe Pro Val Val Glu Ala Asp Leu Ala Leu Phe Gly His Ala His
290 295 300
Leu Gly Thr Gly Asp Pro Tyr Thr Pro Gly Phe Pro Ser Phe Asn His
305 310 315 320
Thr Gln Phe Pro Pro Ser Gln Ser Ser Gly Leu Pro Asn Ile Pro Val
325 330 335
Gln Thr Ile Ser Arg Ala Ala Ala Glu Lys Leu Phe Gly Lys Met Glu
340 345 350
Gly Ser Cys Pro Ala Arg Trp Asn Ile Asp Ser Ser Cys Lys Leu Glu
355 360 365
Leu Ser Gln Asn Gln Asn Val Lys Leu Ile Val Lys Asn Val Leu Lys
370 375 380
Glu Arg Arg Ile Leu Asn Ile Phe Gly Val Ile Lys Gly Tyr Glu Glu
385 390 395 400
Pro Asp Arg Tyr Val Val Val Gly Ala Gln Arg Asp Ala Leu Gly Ala
405 410 415
Gly Val Ala Ala Lys Ser Ser Val Gly Thr Gly Leu Leu Leu Lys Leu
420 425 430
Ala Gln Val Phe Ser Asp Met Ile Ser Lys Asp Gly Phe Arg Pro Ser
435 440 445
Arg Ser Ile Ile Phe Ala Ser Trp Thr Ala Gly Asp Phe Gly Ala Val
450 455 460
Gly Ala Thr Glu Trp Leu Glu Gly Tyr Leu Ser Ser Leu His Leu Lys
465 470 475 480
Ala Phe Thr Tyr Ile Asn Leu Asp Lys Val Val Leu Gly Thr Ser Asn
485 490 495
Phe Lys Val Ser Ala Ser Pro Leu Leu Tyr Thr Leu Met Gly Lys Ile
500 505 510
Met Gln Asp Val Lys His Pro Val Asp Gly Lys Ser Leu Tyr Arg Asp
515 520 525
Ser Asn Trp Ile Ser Lys Val Glu Lys Leu Ser Phe Asp Asn Ala Ala
530 535 540
Tyr Pro Phe Leu Ala Tyr Ser Gly Ile Pro Ala Val Ser Phe Cys Phe
545 550 555 560
Cys Glu Asp Ala Asp Tyr Pro Tyr Leu Gly Thr Arg Leu Asp Thr Tyr
565 570 575
Glu Ala Leu Thr Gln Lys Val Pro Gln Leu Asn Gln Met Val Arg Thr
580 585 590
Ala Ala Glu Val Ala Gly Gln Leu Ile Ile Lys Leu Thr His Asp Val
595 600 605
Glu Leu Asn Leu Asp Tyr Glu Met Tyr Asn Ser Lys Leu Leu Ser Phe
610 615 620
Met Lys Asp Leu Asn Gln Phe Lys Thr Asp Ile Arg Asp Met Gly Leu
625 630 635 640
Ser Leu Gln Trp Leu Tyr Ser Ala Arg Gly Asp Tyr Phe Arg Ala Thr
645 650 655
Ser Arg Leu Thr Thr Asp Phe His Asn Ala Glu Lys Thr Asn Arg Phe
660 665 670
Val Met Arg Glu Ile Asn Asp Arg Ile Met Lys Val Glu Tyr His Phe
675 680 685
Leu Ser Pro Tyr Val Ser Pro Arg Glu Ser Pro Phe Arg His Ile Phe
690 695 700
Trp Gly Ser Gly Ser His Thr Leu Ser Ala Leu Val Glu Asn Leu Lys
705 710 715 720
Leu Arg Gln Lys Asn Ile Thr Ala Phe Asn Glu Thr Leu Phe Arg Asn
725 730 735
Gln Leu Ala Leu Ala Thr Trp Thr Ile Gln Gly Val Ala Asn Ala Leu
740 745 750
Ser Gly Asp Ile Trp Asn Ile Asp Asn Glu Phe
755 760
<210> 88
<211> 672
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of Apical domain mouse chimera of human TfR extracellular
domain
<400> 88
Cys Lys Gly Val Glu Pro Lys Thr Glu Cys Glu Arg Leu Ala Gly Thr
1 5 10 15
Glu Ser Pro Val Arg Glu Glu Pro Gly Glu Asp Phe Pro Ala Ala Arg
20 25 30
Arg Leu Tyr Trp Asp Asp Leu Lys Arg Lys Leu Ser Glu Lys Leu Asp
35 40 45
Ser Thr Asp Phe Thr Gly Thr Ile Lys Leu Leu Asn Glu Asn Ser Tyr
50 55 60
Val Pro Arg Glu Ala Gly Ser Gln Lys Asp Glu Asn Leu Ala Leu Tyr
65 70 75 80
Val Glu Asn Gln Phe Arg Glu Phe Lys Leu Ser Lys Val Trp Arg Asp
85 90 95
Gln His Phe Val Lys Ile Gln Val Lys Ser Ser Ile Gly Gln Asn Met
100 105 110
Val Thr Ile Val Gln Ser Asn Gly Asn Leu Asp Pro Val Glu Ser Pro
115 120 125
Glu Gly Tyr Val Ala Phe Ser Lys Pro Thr Glu Val Ser Gly Lys Leu
130 135 140
Val His Ala Asn Phe Gly Thr Lys Lys Asp Phe Glu Glu Leu Ser Tyr
145 150 155 160
Ser Val Asn Gly Ser Leu Val Ile Val Arg Ala Gly Glu Ile Thr Phe
165 170 175
Ala Glu Lys Val Ala Asn Ala Gln Ser Phe Asn Ala Ile Gly Val Leu
180 185 190
Ile Tyr Met Asp Lys Asn Lys Phe Pro Val Val Glu Ala Asp Leu Ala
195 200 205
Leu Phe Gly His Ala His Leu Gly Thr Gly Asp Pro Tyr Thr Pro Gly
210 215 220
Phe Pro Ser Phe Asn His Thr Gln Phe Pro Pro Ser Gln Ser Ser Gly
225 230 235 240
Leu Pro Asn Ile Pro Val Gln Thr Ile Ser Arg Ala Ala Ala Glu Lys
245 250 255
Leu Phe Gly Lys Met Glu Gly Ser Cys Pro Ala Arg Trp Asn Ile Asp
260 265 270
Ser Ser Cys Lys Leu Glu Leu Ser Gln Asn Gln Asn Val Lys Leu Ile
275 280 285
Val Lys Asn Val Leu Lys Glu Ile Lys Ile Leu Asn Ile Phe Gly Val
290 295 300
Ile Lys Gly Phe Val Glu Pro Asp His Tyr Val Val Val Gly Ala Gln
305 310 315 320
Arg Asp Ala Trp Gly Pro Gly Ala Ala Lys Ser Gly Val Gly Thr Ala
325 330 335
Leu Leu Leu Lys Leu Ala Gln Met Phe Ser Asp Met Val Leu Lys Asp
340 345 350
Gly Phe Gln Pro Ser Arg Ser Ile Ile Phe Ala Ser Trp Ser Ala Gly
355 360 365
Asp Phe Gly Ser Val Gly Ala Thr Glu Trp Leu Glu Gly Tyr Leu Ser
370 375 380
Ser Leu His Leu Lys Ala Phe Thr Tyr Ile Asn Leu Asp Lys Ala Val
385 390 395 400
Leu Gly Thr Ser Asn Phe Lys Val Ser Ala Ser Pro Leu Leu Tyr Thr
405 410 415
Leu Ile Glu Lys Thr Met Gln Asn Val Lys His Pro Val Thr Gly Gln
420 425 430
Phe Leu Tyr Gln Asp Ser Asn Trp Ala Ser Lys Val Glu Lys Leu Thr
435 440 445
Leu Asp Asn Ala Ala Phe Pro Phe Leu Ala Tyr Ser Gly Ile Pro Ala
450 455 460
Val Ser Phe Cys Phe Cys Glu Asp Thr Asp Tyr Pro Tyr Leu Gly Thr
465 470 475 480
Thr Met Asp Thr Tyr Lys Glu Leu Ile Glu Arg Ile Pro Glu Leu Asn
485 490 495
Lys Val Ala Arg Ala Ala Ala Glu Val Ala Gly Gln Phe Val Ile Lys
500 505 510
Leu Thr His Asp Val Glu Leu Asn Leu Asp Tyr Glu Arg Tyr Asn Ser
515 520 525
Gln Leu Leu Ser Phe Val Arg Asp Leu Asn Gln Tyr Arg Ala Asp Ile
530 535 540
Lys Glu Met Gly Leu Ser Leu Gln Trp Leu Tyr Ser Ala Arg Gly Asp
545 550 555 560
Phe Phe Arg Ala Thr Ser Arg Leu Thr Thr Asp Phe Gly Asn Ala Glu
565 570 575
Lys Thr Asp Arg Phe Val Met Lys Lys Leu Asn Asp Arg Val Met Arg
580 585 590
Val Glu Tyr His Phe Leu Ser Pro Tyr Val Ser Pro Lys Glu Ser Pro
595 600 605
Phe Arg His Val Phe Trp Gly Ser Gly Ser His Thr Leu Pro Ala Leu
610 615 620
Leu Glu Asn Leu Lys Leu Arg Lys Gln Asn Asn Gly Ala Phe Asn Glu
625 630 635 640
Thr Leu Phe Arg Asn Gln Leu Ala Leu Ala Thr Trp Thr Ile Gln Gly
645 650 655
Ala Ala Asn Ala Leu Ser Gly Asp Val Trp Asp Ile Asp Asn Glu Phe
660 665 670
<210> 89
<211> 675
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of Protease-like domain mouse chimera of human TfR
extracellular
domain
<400> 89
Cys Lys Arg Val Glu Gln Lys Glu Glu Cys Val Lys Leu Ala Glu Thr
1 5 10 15
Glu Glu Thr Asp Lys Ser Glu Thr Met Glu Thr Glu Asp Val Pro Thr
20 25 30
Ser Ser Arg Leu Tyr Trp Ala Asp Leu Lys Thr Leu Leu Ser Glu Lys
35 40 45
Leu Asn Ser Ile Glu Phe Ala Asp Thr Ile Lys Gln Leu Ser Gln Asn
50 55 60
Thr Tyr Thr Pro Arg Glu Ala Gly Ser Gln Lys Asp Glu Ser Leu Ala
65 70 75 80
Tyr Tyr Ile Glu Asn Gln Phe His Glu Phe Lys Phe Ser Lys Val Trp
85 90 95
Arg Asp Glu His Tyr Val Lys Ile Gln Val Lys Asp Ser Ala Gln Asn
100 105 110
Ser Val Ile Ile Val Asp Lys Asn Gly Arg Leu Val Tyr Leu Val Glu
115 120 125
Asn Pro Gly Gly Tyr Val Ala Tyr Ser Lys Ala Ala Thr Val Thr Gly
130 135 140
Lys Leu Val His Ala Asn Phe Gly Thr Lys Lys Asp Phe Glu Asp Leu
145 150 155 160
Tyr Thr Pro Val Asn Gly Ser Ile Val Ile Val Arg Ala Gly Lys Ile
165 170 175
Thr Phe Ala Glu Lys Val Ala Asn Ala Glu Ser Leu Asn Ala Ile Gly
180 185 190
Val Leu Ile Tyr Met Asp Gln Thr Lys Phe Pro Ile Val Asn Ala Glu
195 200 205
Leu Ser Phe Phe Gly His Ala His Leu Gly Thr Gly Asp Pro Tyr Thr
210 215 220
Pro Gly Phe Pro Ser Phe Asn His Thr Gln Phe Pro Pro Ser Arg Ser
225 230 235 240
Ser Gly Leu Pro Asn Ile Pro Val Gln Thr Ile Ser Arg Ala Ala Ala
245 250 255
Glu Lys Leu Phe Gly Asn Met Glu Gly Asp Cys Pro Ser Asp Trp Lys
260 265 270
Thr Asp Ser Thr Cys Arg Met Val Thr Ser Glu Ser Lys Asn Val Lys
275 280 285
Leu Thr Val Ser Asn Val Leu Lys Glu Arg Arg Ile Leu Asn Ile Phe
290 295 300
Gly Val Ile Lys Gly Tyr Glu Glu Pro Asp Arg Tyr Val Val Val Gly
305 310 315 320
Ala Gln Arg Asp Ala Leu Gly Ala Gly Val Ala Ala Lys Ser Ser Val
325 330 335
Gly Thr Gly Leu Leu Leu Lys Leu Ala Gln Val Phe Ser Asp Met Ile
340 345 350
Ser Lys Asp Gly Phe Arg Pro Ser Arg Ser Ile Ile Phe Ala Ser Trp
355 360 365
Thr Ala Gly Asp Phe Gly Ala Val Gly Ala Thr Glu Trp Leu Glu Gly
370 375 380
Tyr Leu Ser Ser Leu His Leu Lys Ala Phe Thr Tyr Ile Asn Leu Asp
385 390 395 400
Lys Val Val Leu Gly Thr Ser Asn Phe Lys Val Ser Ala Ser Pro Leu
405 410 415
Leu Tyr Thr Leu Met Gly Lys Ile Met Gln Asp Val Lys His Pro Val
420 425 430
Asp Gly Lys Ser Leu Tyr Arg Asp Ser Asn Trp Ile Ser Lys Val Glu
435 440 445
Lys Leu Ser Phe Asp Asn Ala Ala Tyr Pro Phe Leu Ala Tyr Ser Gly
450 455 460
Ile Pro Ala Val Ser Phe Cys Phe Cys Glu Asp Ala Asp Tyr Pro Tyr
465 470 475 480
Leu Gly Thr Arg Leu Asp Thr Tyr Glu Ala Leu Thr Gln Lys Val Pro
485 490 495
Gln Leu Asn Gln Met Val Arg Thr Ala Ala Glu Val Ala Gly Gln Leu
500 505 510
Ile Ile Lys Leu Thr His Asp Val Glu Leu Asn Leu Asp Tyr Glu Arg
515 520 525
Tyr Asn Ser Gln Leu Leu Ser Phe Val Arg Asp Leu Asn Gln Tyr Arg
530 535 540
Ala Asp Ile Lys Glu Met Gly Leu Ser Leu Gln Trp Leu Tyr Ser Ala
545 550 555 560
Arg Gly Asp Phe Phe Arg Ala Thr Ser Arg Leu Thr Thr Asp Phe Gly
565 570 575
Asn Ala Glu Lys Thr Asp Arg Phe Val Met Lys Lys Leu Asn Asp Arg
580 585 590
Val Met Arg Val Glu Tyr His Phe Leu Ser Pro Tyr Val Ser Pro Lys
595 600 605
Glu Ser Pro Phe Arg His Val Phe Trp Gly Ser Gly Ser His Thr Leu
610 615 620
Pro Ala Leu Leu Glu Asn Leu Lys Leu Arg Lys Gln Asn Asn Gly Ala
625 630 635 640
Phe Asn Glu Thr Leu Phe Arg Asn Gln Leu Ala Leu Ala Thr Trp Thr
645 650 655
Ile Gln Gly Ala Ala Asn Ala Leu Ser Gly Asp Val Trp Asp Ile Asp
660 665 670
Asn Glu Phe
675
<210> 90
<211> 672
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of A01
<400> 90
Cys Lys Gly Val Glu Pro Lys Thr Glu Cys Glu Arg Leu Ala Gly Thr
1 5 10 15
Glu Ser Pro Val Arg Glu Glu Pro Gly Glu Asp Phe Pro Ala Ala Arg
20 25 30
Arg Leu Tyr Trp Asp Asp Leu Lys Arg Lys Leu Ser Glu Lys Leu Asp
35 40 45
Ser Thr Asp Phe Thr Gly Thr Ile Lys Leu Leu Asn Glu Asn Ser Tyr
50 55 60
Val Pro Arg Glu Ala Gly Ser Gln Lys Asp Glu Asn Leu Ala Leu Tyr
65 70 75 80
Val Glu Asn Gln Phe Arg Glu Phe Lys Leu Ser Lys Val Trp Arg Asp
85 90 95
Gln His Phe Val Lys Ile Gln Val Lys Asp Ser Ala Gln Asn Ser Val
100 105 110
Ile Ile Val Asp Lys Asn Gly Arg Leu Val Tyr Leu Val Glu Asn Pro
115 120 125
Gly Gly Tyr Val Ala Tyr Ser Lys Ala Ala Thr Val Thr Gly Lys Leu
130 135 140
Val His Ala Asn Phe Gly Thr Lys Lys Asp Phe Glu Asp Leu Tyr Thr
145 150 155 160
Pro Val Asn Gly Ser Ile Val Ile Val Arg Ala Gly Lys Ile Thr Phe
165 170 175
Ala Glu Lys Val Ala Asn Ala Glu Ser Leu Asn Ala Ile Gly Val Leu
180 185 190
Ile Tyr Met Asp Lys Asn Lys Phe Pro Ile Val Asn Ala Glu Leu Ser
195 200 205
Phe Phe Gly His Ala His Leu Gly Thr Gly Asp Pro Tyr Thr Pro Gly
210 215 220
Phe Pro Ser Phe Asn His Thr Gln Phe Pro Pro Ser Arg Ser Ser Gly
225 230 235 240
Leu Pro Asn Ile Pro Val Gln Thr Ile Ser Arg Ala Ala Ala Glu Lys
245 250 255
Leu Phe Gly Asn Met Glu Gly Asp Cys Pro Ser Asp Trp Lys Thr Asp
260 265 270
Ser Ser Cys Arg Met Val Thr Ser Glu Ser Lys Asn Val Lys Leu Thr
275 280 285
Val Ser Asn Val Leu Lys Glu Ile Lys Ile Leu Asn Ile Phe Gly Val
290 295 300
Ile Lys Gly Phe Val Glu Pro Asp His Tyr Val Val Val Gly Ala Gln
305 310 315 320
Arg Asp Ala Trp Gly Pro Gly Ala Ala Lys Ser Gly Val Gly Thr Ala
325 330 335
Leu Leu Leu Lys Leu Ala Gln Met Phe Ser Asp Met Val Leu Lys Asp
340 345 350
Gly Phe Gln Pro Ser Arg Ser Ile Ile Phe Ala Ser Trp Ser Ala Gly
355 360 365
Asp Phe Gly Ser Val Gly Ala Thr Glu Trp Leu Glu Gly Tyr Leu Ser
370 375 380
Ser Leu His Leu Lys Ala Phe Thr Tyr Ile Asn Leu Asp Lys Ala Val
385 390 395 400
Leu Gly Thr Ser Asn Phe Lys Val Ser Ala Ser Pro Leu Leu Tyr Thr
405 410 415
Leu Ile Glu Lys Thr Met Gln Asn Val Lys His Pro Val Thr Gly Gln
420 425 430
Phe Leu Tyr Gln Asp Ser Asn Trp Ala Ser Lys Val Glu Lys Leu Thr
435 440 445
Leu Asp Asn Ala Ala Phe Pro Phe Leu Ala Tyr Ser Gly Ile Pro Ala
450 455 460
Val Ser Phe Cys Phe Cys Glu Asp Thr Asp Tyr Pro Tyr Leu Gly Thr
465 470 475 480
Thr Met Asp Thr Tyr Lys Glu Leu Ile Glu Arg Ile Pro Glu Leu Asn
485 490 495
Lys Val Ala Arg Ala Ala Ala Glu Val Ala Gly Gln Phe Val Ile Lys
500 505 510
Leu Thr His Asp Val Glu Leu Asn Leu Asp Tyr Glu Arg Tyr Asn Ser
515 520 525
Gln Leu Leu Ser Phe Val Arg Asp Leu Asn Gln Tyr Arg Ala Asp Ile
530 535 540
Lys Glu Met Gly Leu Ser Leu Gln Trp Leu Tyr Ser Ala Arg Gly Asp
545 550 555 560
Phe Phe Arg Ala Thr Ser Arg Leu Thr Thr Asp Phe Gly Asn Ala Glu
565 570 575
Lys Thr Asp Arg Phe Val Met Lys Lys Leu Asn Asp Arg Val Met Arg
580 585 590
Val Glu Tyr His Phe Leu Ser Pro Tyr Val Ser Pro Lys Glu Ser Pro
595 600 605
Phe Arg His Val Phe Trp Gly Ser Gly Ser His Thr Leu Pro Ala Leu
610 615 620
Leu Glu Asn Leu Lys Leu Arg Lys Gln Asn Asn Gly Ala Phe Asn Glu
625 630 635 640
Thr Leu Phe Arg Asn Gln Leu Ala Leu Ala Thr Trp Thr Ile Gln Gly
645 650 655
Ala Ala Asn Ala Leu Ser Gly Asp Val Trp Asp Ile Asp Asn Glu Phe
660 665 670
<210> 91
<211> 672
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of A02
<400> 91
Cys Lys Gly Val Glu Pro Lys Thr Glu Cys Glu Arg Leu Ala Gly Thr
1 5 10 15
Glu Ser Pro Val Arg Glu Glu Pro Gly Glu Asp Phe Pro Ala Ala Arg
20 25 30
Arg Leu Tyr Trp Asp Asp Leu Lys Arg Lys Leu Ser Glu Lys Leu Asp
35 40 45
Ser Thr Asp Phe Thr Gly Thr Ile Lys Leu Leu Asn Glu Asn Ser Tyr
50 55 60
Val Pro Arg Glu Ala Gly Ser Gln Lys Asp Glu Asn Leu Ala Leu Tyr
65 70 75 80
Val Glu Asn Gln Phe Arg Glu Phe Lys Leu Ser Lys Val Trp Arg Asp
85 90 95
Gln His Phe Val Lys Ile Gln Val Lys Asp Ser Ala Gln Asn Ser Val
100 105 110
Ile Ile Val Asp Lys Asn Gly Arg Leu Val Tyr Leu Val Glu Asn Pro
115 120 125
Gly Gly Tyr Val Ala Tyr Ser Lys Ala Ala Thr Val Thr Gly Lys Leu
130 135 140
Val His Ala Asn Phe Gly Thr Lys Lys Asp Phe Glu Asp Leu Tyr Thr
145 150 155 160
Pro Val Asn Gly Ser Ile Val Ile Val Arg Ala Gly Lys Ile Thr Phe
165 170 175
Ala Glu Lys Val Ala Asn Ala Glu Ser Leu Asn Ala Ile Gly Val Leu
180 185 190
Ile Tyr Met Asp Gln Thr Lys Phe Pro Ile Val Asn Ala Glu Leu Ser
195 200 205
Phe Phe Gly His Ala His Leu Gly Thr Gly Asp Pro Tyr Thr Pro Gly
210 215 220
Phe Pro Ser Phe Asn His Thr Gln Phe Pro Pro Ser Arg Ser Ser Gly
225 230 235 240
Leu Pro Asn Ile Pro Val Gln Thr Ile Ser Arg Ala Ala Ala Glu Lys
245 250 255
Leu Phe Gly Asn Met Glu Gly Ser Cys Pro Ala Arg Trp Asn Thr Asp
260 265 270
Ser Thr Cys Arg Met Val Thr Ser Glu Ser Lys Asn Val Lys Leu Thr
275 280 285
Val Ser Asn Val Leu Lys Glu Ile Lys Ile Leu Asn Ile Phe Gly Val
290 295 300
Ile Lys Gly Phe Val Glu Pro Asp His Tyr Val Val Val Gly Ala Gln
305 310 315 320
Arg Asp Ala Trp Gly Pro Gly Ala Ala Lys Ser Gly Val Gly Thr Ala
325 330 335
Leu Leu Leu Lys Leu Ala Gln Met Phe Ser Asp Met Val Leu Lys Asp
340 345 350
Gly Phe Gln Pro Ser Arg Ser Ile Ile Phe Ala Ser Trp Ser Ala Gly
355 360 365
Asp Phe Gly Ser Val Gly Ala Thr Glu Trp Leu Glu Gly Tyr Leu Ser
370 375 380
Ser Leu His Leu Lys Ala Phe Thr Tyr Ile Asn Leu Asp Lys Ala Val
385 390 395 400
Leu Gly Thr Ser Asn Phe Lys Val Ser Ala Ser Pro Leu Leu Tyr Thr
405 410 415
Leu Ile Glu Lys Thr Met Gln Asn Val Lys His Pro Val Thr Gly Gln
420 425 430
Phe Leu Tyr Gln Asp Ser Asn Trp Ala Ser Lys Val Glu Lys Leu Thr
435 440 445
Leu Asp Asn Ala Ala Phe Pro Phe Leu Ala Tyr Ser Gly Ile Pro Ala
450 455 460
Val Ser Phe Cys Phe Cys Glu Asp Thr Asp Tyr Pro Tyr Leu Gly Thr
465 470 475 480
Thr Met Asp Thr Tyr Lys Glu Leu Ile Glu Arg Ile Pro Glu Leu Asn
485 490 495
Lys Val Ala Arg Ala Ala Ala Glu Val Ala Gly Gln Phe Val Ile Lys
500 505 510
Leu Thr His Asp Val Glu Leu Asn Leu Asp Tyr Glu Arg Tyr Asn Ser
515 520 525
Gln Leu Leu Ser Phe Val Arg Asp Leu Asn Gln Tyr Arg Ala Asp Ile
530 535 540
Lys Glu Met Gly Leu Ser Leu Gln Trp Leu Tyr Ser Ala Arg Gly Asp
545 550 555 560
Phe Phe Arg Ala Thr Ser Arg Leu Thr Thr Asp Phe Gly Asn Ala Glu
565 570 575
Lys Thr Asp Arg Phe Val Met Lys Lys Leu Asn Asp Arg Val Met Arg
580 585 590
Val Glu Tyr His Phe Leu Ser Pro Tyr Val Ser Pro Lys Glu Ser Pro
595 600 605
Phe Arg His Val Phe Trp Gly Ser Gly Ser His Thr Leu Pro Ala Leu
610 615 620
Leu Glu Asn Leu Lys Leu Arg Lys Gln Asn Asn Gly Ala Phe Asn Glu
625 630 635 640
Thr Leu Phe Arg Asn Gln Leu Ala Leu Ala Thr Trp Thr Ile Gln Gly
645 650 655
Ala Ala Asn Ala Leu Ser Gly Asp Val Trp Asp Ile Asp Asn Glu Phe
660 665 670
<210> 92
<211> 672
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of A03
<400> 92
Cys Lys Gly Val Glu Pro Lys Thr Glu Cys Glu Arg Leu Ala Gly Thr
1 5 10 15
Glu Ser Pro Val Arg Glu Glu Pro Gly Glu Asp Phe Pro Ala Ala Arg
20 25 30
Arg Leu Tyr Trp Asp Asp Leu Lys Arg Lys Leu Ser Glu Lys Leu Asp
35 40 45
Ser Thr Asp Phe Thr Gly Thr Ile Lys Leu Leu Asn Glu Asn Ser Tyr
50 55 60
Val Pro Arg Glu Ala Gly Ser Gln Lys Asp Glu Asn Leu Ala Leu Tyr
65 70 75 80
Val Glu Asn Gln Phe Arg Glu Phe Lys Leu Ser Lys Val Trp Arg Asp
85 90 95
Gln His Phe Val Lys Ile Gln Val Lys Asp Ser Ala Gln Asn Ser Val
100 105 110
Ile Ile Val Asp Lys Asn Gly Arg Leu Val Tyr Leu Val Glu Asn Pro
115 120 125
Gly Gly Tyr Val Ala Tyr Ser Lys Ala Ala Thr Val Thr Gly Lys Leu
130 135 140
Val His Ala Asn Phe Gly Thr Lys Lys Asp Phe Glu Glu Leu Tyr Thr
145 150 155 160
Pro Val Asn Gly Ser Ile Val Ile Val Arg Ala Gly Glu Ile Thr Phe
165 170 175
Ala Glu Lys Val Ala Asn Ala Glu Ser Leu Asn Ala Ile Gly Val Leu
180 185 190
Ile Tyr Met Asp Gln Thr Lys Phe Pro Ile Val Asn Ala Glu Leu Ser
195 200 205
Phe Phe Gly His Ala His Leu Gly Thr Gly Asp Pro Tyr Thr Pro Gly
210 215 220
Phe Pro Ser Phe Asn His Thr Gln Phe Pro Pro Ser Arg Ser Ser Gly
225 230 235 240
Leu Pro Asn Ile Pro Val Gln Thr Ile Ser Arg Ala Ala Ala Glu Lys
245 250 255
Leu Phe Gly Asn Met Glu Gly Asp Cys Pro Ser Asp Trp Lys Thr Asp
260 265 270
Ser Thr Cys Arg Met Val Thr Ser Glu Ser Lys Asn Val Lys Leu Thr
275 280 285
Val Ser Asn Val Leu Lys Glu Ile Lys Ile Leu Asn Ile Phe Gly Val
290 295 300
Ile Lys Gly Phe Val Glu Pro Asp His Tyr Val Val Val Gly Ala Gln
305 310 315 320
Arg Asp Ala Trp Gly Pro Gly Ala Ala Lys Ser Gly Val Gly Thr Ala
325 330 335
Leu Leu Leu Lys Leu Ala Gln Met Phe Ser Asp Met Val Leu Lys Asp
340 345 350
Gly Phe Gln Pro Ser Arg Ser Ile Ile Phe Ala Ser Trp Ser Ala Gly
355 360 365
Asp Phe Gly Ser Val Gly Ala Thr Glu Trp Leu Glu Gly Tyr Leu Ser
370 375 380
Ser Leu His Leu Lys Ala Phe Thr Tyr Ile Asn Leu Asp Lys Ala Val
385 390 395 400
Leu Gly Thr Ser Asn Phe Lys Val Ser Ala Ser Pro Leu Leu Tyr Thr
405 410 415
Leu Ile Glu Lys Thr Met Gln Asn Val Lys His Pro Val Thr Gly Gln
420 425 430
Phe Leu Tyr Gln Asp Ser Asn Trp Ala Ser Lys Val Glu Lys Leu Thr
435 440 445
Leu Asp Asn Ala Ala Phe Pro Phe Leu Ala Tyr Ser Gly Ile Pro Ala
450 455 460
Val Ser Phe Cys Phe Cys Glu Asp Thr Asp Tyr Pro Tyr Leu Gly Thr
465 470 475 480
Thr Met Asp Thr Tyr Lys Glu Leu Ile Glu Arg Ile Pro Glu Leu Asn
485 490 495
Lys Val Ala Arg Ala Ala Ala Glu Val Ala Gly Gln Phe Val Ile Lys
500 505 510
Leu Thr His Asp Val Glu Leu Asn Leu Asp Tyr Glu Arg Tyr Asn Ser
515 520 525
Gln Leu Leu Ser Phe Val Arg Asp Leu Asn Gln Tyr Arg Ala Asp Ile
530 535 540
Lys Glu Met Gly Leu Ser Leu Gln Trp Leu Tyr Ser Ala Arg Gly Asp
545 550 555 560
Phe Phe Arg Ala Thr Ser Arg Leu Thr Thr Asp Phe Gly Asn Ala Glu
565 570 575
Lys Thr Asp Arg Phe Val Met Lys Lys Leu Asn Asp Arg Val Met Arg
580 585 590
Val Glu Tyr His Phe Leu Ser Pro Tyr Val Ser Pro Lys Glu Ser Pro
595 600 605
Phe Arg His Val Phe Trp Gly Ser Gly Ser His Thr Leu Pro Ala Leu
610 615 620
Leu Glu Asn Leu Lys Leu Arg Lys Gln Asn Asn Gly Ala Phe Asn Glu
625 630 635 640
Thr Leu Phe Arg Asn Gln Leu Ala Leu Ala Thr Trp Thr Ile Gln Gly
645 650 655
Ala Ala Asn Ala Leu Ser Gly Asp Val Trp Asp Ile Asp Asn Glu Phe
660 665 670
<210> 93
<211> 672
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of A04
<400> 93
Cys Lys Gly Val Glu Pro Lys Thr Glu Cys Glu Arg Leu Ala Gly Thr
1 5 10 15
Glu Ser Pro Val Arg Glu Glu Pro Gly Glu Asp Phe Pro Ala Ala Arg
20 25 30
Arg Leu Tyr Trp Asp Asp Leu Lys Arg Lys Leu Ser Glu Lys Leu Asp
35 40 45
Ser Thr Asp Phe Thr Gly Thr Ile Lys Leu Leu Asn Glu Asn Ser Tyr
50 55 60
Val Pro Arg Glu Ala Gly Ser Gln Lys Asp Glu Asn Leu Ala Leu Tyr
65 70 75 80
Val Glu Asn Gln Phe Arg Glu Phe Lys Leu Ser Lys Val Trp Arg Asp
85 90 95
Gln His Phe Val Lys Ile Gln Val Lys Asp Ser Ala Gln Asn Ser Val
100 105 110
Ile Ile Val Asp Lys Asn Gly Arg Leu Val Tyr Leu Val Glu Asn Pro
115 120 125
Gly Gly Tyr Val Ala Tyr Ser Lys Ala Ala Thr Val Thr Gly Lys Leu
130 135 140
Val His Ala Asn Phe Gly Thr Lys Lys Asp Phe Glu Glu Leu Tyr Thr
145 150 155 160
Pro Val Asn Gly Ser Ile Val Ile Val Arg Ala Gly Glu Ile Thr Phe
165 170 175
Ala Glu Lys Val Ala Asn Ala Glu Ser Leu Asn Ala Ile Gly Val Leu
180 185 190
Ile Tyr Met Asp Gln Thr Lys Phe Pro Ile Val Asn Ala Glu Leu Ser
195 200 205
Phe Phe Gly His Ala His Leu Gly Thr Gly Asp Pro Tyr Thr Pro Gly
210 215 220
Phe Pro Ser Phe Asn His Thr Gln Phe Pro Pro Ser Arg Ser Ser Gly
225 230 235 240
Leu Pro Asn Ile Pro Val Gln Thr Ile Ser Arg Ala Ala Ala Glu Lys
245 250 255
Leu Phe Gly Asn Met Glu Gly Asp Cys Pro Ser Arg Trp Lys Thr Asp
260 265 270
Ser Thr Cys Arg Met Val Thr Ser Glu Ser Lys Asn Val Lys Leu Thr
275 280 285
Val Ser Asn Val Leu Lys Glu Ile Lys Ile Leu Asn Ile Phe Gly Val
290 295 300
Ile Lys Gly Phe Val Glu Pro Asp His Tyr Val Val Val Gly Ala Gln
305 310 315 320
Arg Asp Ala Trp Gly Pro Gly Ala Ala Lys Ser Gly Val Gly Thr Ala
325 330 335
Leu Leu Leu Lys Leu Ala Gln Met Phe Ser Asp Met Val Leu Lys Asp
340 345 350
Gly Phe Gln Pro Ser Arg Ser Ile Ile Phe Ala Ser Trp Ser Ala Gly
355 360 365
Asp Phe Gly Ser Val Gly Ala Thr Glu Trp Leu Glu Gly Tyr Leu Ser
370 375 380
Ser Leu His Leu Lys Ala Phe Thr Tyr Ile Asn Leu Asp Lys Ala Val
385 390 395 400
Leu Gly Thr Ser Asn Phe Lys Val Ser Ala Ser Pro Leu Leu Tyr Thr
405 410 415
Leu Ile Glu Lys Thr Met Gln Asn Val Lys His Pro Val Thr Gly Gln
420 425 430
Phe Leu Tyr Gln Asp Ser Asn Trp Ala Ser Lys Val Glu Lys Leu Thr
435 440 445
Leu Asp Asn Ala Ala Phe Pro Phe Leu Ala Tyr Ser Gly Ile Pro Ala
450 455 460
Val Ser Phe Cys Phe Cys Glu Asp Thr Asp Tyr Pro Tyr Leu Gly Thr
465 470 475 480
Thr Met Asp Thr Tyr Lys Glu Leu Ile Glu Arg Ile Pro Glu Leu Asn
485 490 495
Lys Val Ala Arg Ala Ala Ala Glu Val Ala Gly Gln Phe Val Ile Lys
500 505 510
Leu Thr His Asp Val Glu Leu Asn Leu Asp Tyr Glu Arg Tyr Asn Ser
515 520 525
Gln Leu Leu Ser Phe Val Arg Asp Leu Asn Gln Tyr Arg Ala Asp Ile
530 535 540
Lys Glu Met Gly Leu Ser Leu Gln Trp Leu Tyr Ser Ala Arg Gly Asp
545 550 555 560
Phe Phe Arg Ala Thr Ser Arg Leu Thr Thr Asp Phe Gly Asn Ala Glu
565 570 575
Lys Thr Asp Arg Phe Val Met Lys Lys Leu Asn Asp Arg Val Met Arg
580 585 590
Val Glu Tyr His Phe Leu Ser Pro Tyr Val Ser Pro Lys Glu Ser Pro
595 600 605
Phe Arg His Val Phe Trp Gly Ser Gly Ser His Thr Leu Pro Ala Leu
610 615 620
Leu Glu Asn Leu Lys Leu Arg Lys Gln Asn Asn Gly Ala Phe Asn Glu
625 630 635 640
Thr Leu Phe Arg Asn Gln Leu Ala Leu Ala Thr Trp Thr Ile Gln Gly
645 650 655
Ala Ala Asn Ala Leu Ser Gly Asp Val Trp Asp Ile Asp Asn Glu Phe
660 665 670
<210> 94
<211> 672
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of A05
<400> 94
Cys Lys Gly Val Glu Pro Lys Thr Glu Cys Glu Arg Leu Ala Gly Thr
1 5 10 15
Glu Ser Pro Val Arg Glu Glu Pro Gly Glu Asp Phe Pro Ala Ala Arg
20 25 30
Arg Leu Tyr Trp Asp Asp Leu Lys Arg Lys Leu Ser Glu Lys Leu Asp
35 40 45
Ser Thr Asp Phe Thr Gly Thr Ile Lys Leu Leu Asn Glu Asn Ser Tyr
50 55 60
Val Pro Arg Glu Ala Gly Ser Gln Lys Asp Glu Asn Leu Ala Leu Tyr
65 70 75 80
Val Glu Asn Gln Phe Arg Glu Phe Lys Leu Ser Lys Val Trp Arg Asp
85 90 95
Gln His Phe Val Lys Ile Gln Val Lys Asp Ser Ala Gln Asn Ser Val
100 105 110
Ile Ile Val Asp Lys Asn Gly Arg Leu Val Tyr Leu Val Glu Asn Pro
115 120 125
Gly Gly Tyr Val Ala Tyr Ser Lys Ala Ala Thr Val Thr Gly Lys Leu
130 135 140
Val His Ala Asn Phe Gly Thr Lys Lys Asp Phe Glu Asp Leu Tyr Thr
145 150 155 160
Ser Val Asn Gly Ser Ile Val Ile Val Arg Ala Gly Lys Ile Thr Phe
165 170 175
Ala Glu Lys Val Ala Asn Ala Glu Ser Phe Asn Ala Ile Gly Val Leu
180 185 190
Ile Tyr Met Asp Gln Thr Lys Phe Pro Ile Val Asn Ala Glu Leu Ser
195 200 205
Phe Phe Gly His Ala His Leu Gly Thr Gly Asp Pro Tyr Thr Pro Gly
210 215 220
Phe Pro Ser Phe Asn His Thr Gln Phe Pro Pro Ser Arg Ser Ser Gly
225 230 235 240
Leu Pro Asn Ile Pro Val Gln Thr Ile Ser Arg Ala Ala Ala Glu Lys
245 250 255
Leu Phe Gly Asn Met Glu Gly Asp Cys Pro Ser Asp Trp Lys Thr Asp
260 265 270
Ser Thr Cys Arg Met Val Thr Ser Glu Ser Lys Asn Val Lys Leu Thr
275 280 285
Val Ser Asn Val Leu Lys Glu Ile Lys Ile Leu Asn Ile Phe Gly Val
290 295 300
Ile Lys Gly Phe Val Glu Pro Asp His Tyr Val Val Val Gly Ala Gln
305 310 315 320
Arg Asp Ala Trp Gly Pro Gly Ala Ala Lys Ser Gly Val Gly Thr Ala
325 330 335
Leu Leu Leu Lys Leu Ala Gln Met Phe Ser Asp Met Val Leu Lys Asp
340 345 350
Gly Phe Gln Pro Ser Arg Ser Ile Ile Phe Ala Ser Trp Ser Ala Gly
355 360 365
Asp Phe Gly Ser Val Gly Ala Thr Glu Trp Leu Glu Gly Tyr Leu Ser
370 375 380
Ser Leu His Leu Lys Ala Phe Thr Tyr Ile Asn Leu Asp Lys Ala Val
385 390 395 400
Leu Gly Thr Ser Asn Phe Lys Val Ser Ala Ser Pro Leu Leu Tyr Thr
405 410 415
Leu Ile Glu Lys Thr Met Gln Asn Val Lys His Pro Val Thr Gly Gln
420 425 430
Phe Leu Tyr Gln Asp Ser Asn Trp Ala Ser Lys Val Glu Lys Leu Thr
435 440 445
Leu Asp Asn Ala Ala Phe Pro Phe Leu Ala Tyr Ser Gly Ile Pro Ala
450 455 460
Val Ser Phe Cys Phe Cys Glu Asp Thr Asp Tyr Pro Tyr Leu Gly Thr
465 470 475 480
Thr Met Asp Thr Tyr Lys Glu Leu Ile Glu Arg Ile Pro Glu Leu Asn
485 490 495
Lys Val Ala Arg Ala Ala Ala Glu Val Ala Gly Gln Phe Val Ile Lys
500 505 510
Leu Thr His Asp Val Glu Leu Asn Leu Asp Tyr Glu Arg Tyr Asn Ser
515 520 525
Gln Leu Leu Ser Phe Val Arg Asp Leu Asn Gln Tyr Arg Ala Asp Ile
530 535 540
Lys Glu Met Gly Leu Ser Leu Gln Trp Leu Tyr Ser Ala Arg Gly Asp
545 550 555 560
Phe Phe Arg Ala Thr Ser Arg Leu Thr Thr Asp Phe Gly Asn Ala Glu
565 570 575
Lys Thr Asp Arg Phe Val Met Lys Lys Leu Asn Asp Arg Val Met Arg
580 585 590
Val Glu Tyr His Phe Leu Ser Pro Tyr Val Ser Pro Lys Glu Ser Pro
595 600 605
Phe Arg His Val Phe Trp Gly Ser Gly Ser His Thr Leu Pro Ala Leu
610 615 620
Leu Glu Asn Leu Lys Leu Arg Lys Gln Asn Asn Gly Ala Phe Asn Glu
625 630 635 640
Thr Leu Phe Arg Asn Gln Leu Ala Leu Ala Thr Trp Thr Ile Gln Gly
645 650 655
Ala Ala Asn Ala Leu Ser Gly Asp Val Trp Asp Ile Asp Asn Glu Phe
660 665 670
<210> 95
<211> 672
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of A06
<400> 95
Cys Lys Gly Val Glu Pro Lys Thr Glu Cys Glu Arg Leu Ala Gly Thr
1 5 10 15
Glu Ser Pro Val Arg Glu Glu Pro Gly Glu Asp Phe Pro Ala Ala Arg
20 25 30
Arg Leu Tyr Trp Asp Asp Leu Lys Arg Lys Leu Ser Glu Lys Leu Asp
35 40 45
Ser Thr Asp Phe Thr Gly Thr Ile Lys Leu Leu Asn Glu Asn Ser Tyr
50 55 60
Val Pro Arg Glu Ala Gly Ser Gln Lys Asp Glu Asn Leu Ala Leu Tyr
65 70 75 80
Val Glu Asn Gln Phe Arg Glu Phe Lys Leu Ser Lys Val Trp Arg Asp
85 90 95
Gln His Phe Val Lys Ile Gln Val Lys Asp Ser Ala Gln Asn Ser Val
100 105 110
Ile Ile Val Asp Lys Asn Gly Arg Leu Val Tyr Leu Val Glu Asn Pro
115 120 125
Gly Gly Tyr Val Ala Tyr Ser Lys Ala Ala Thr Val Thr Gly Lys Leu
130 135 140
Val His Ala Asn Phe Gly Thr Lys Lys Asp Phe Glu Asp Leu Tyr Thr
145 150 155 160
Pro Val Asn Gly Ser Ile Val Ile Val Arg Ala Gly Lys Ile Thr Phe
165 170 175
Ala Glu Lys Val Ala Asn Ala Gln Ser Leu Asn Ala Ile Gly Val Leu
180 185 190
Ile Tyr Met Asp Gln Thr Lys Phe Pro Ile Val Asn Ala Glu Leu Ser
195 200 205
Phe Phe Gly His Ala His Leu Gly Thr Gly Asp Pro Tyr Thr Pro Gly
210 215 220
Phe Pro Ser Phe Asn His Thr Gln Phe Pro Pro Ser Arg Ser Ser Gly
225 230 235 240
Leu Pro Asn Ile Pro Val Gln Thr Ile Ser Arg Ala Ala Ala Glu Lys
245 250 255
Leu Phe Gly Asn Met Glu Gly Asp Cys Pro Ser Asp Trp Lys Thr Asp
260 265 270
Ser Thr Cys Arg Met Val Thr Ser Glu Ser Lys Asn Val Lys Leu Thr
275 280 285
Val Ser Asn Val Leu Lys Glu Ile Lys Ile Leu Asn Ile Phe Gly Val
290 295 300
Ile Lys Gly Phe Val Glu Pro Asp His Tyr Val Val Val Gly Ala Gln
305 310 315 320
Arg Asp Ala Trp Gly Pro Gly Ala Ala Lys Ser Gly Val Gly Thr Ala
325 330 335
Leu Leu Leu Lys Leu Ala Gln Met Phe Ser Asp Met Val Leu Lys Asp
340 345 350
Gly Phe Gln Pro Ser Arg Ser Ile Ile Phe Ala Ser Trp Ser Ala Gly
355 360 365
Asp Phe Gly Ser Val Gly Ala Thr Glu Trp Leu Glu Gly Tyr Leu Ser
370 375 380
Ser Leu His Leu Lys Ala Phe Thr Tyr Ile Asn Leu Asp Lys Ala Val
385 390 395 400
Leu Gly Thr Ser Asn Phe Lys Val Ser Ala Ser Pro Leu Leu Tyr Thr
405 410 415
Leu Ile Glu Lys Thr Met Gln Asn Val Lys His Pro Val Thr Gly Gln
420 425 430
Phe Leu Tyr Gln Asp Ser Asn Trp Ala Ser Lys Val Glu Lys Leu Thr
435 440 445
Leu Asp Asn Ala Ala Phe Pro Phe Leu Ala Tyr Ser Gly Ile Pro Ala
450 455 460
Val Ser Phe Cys Phe Cys Glu Asp Thr Asp Tyr Pro Tyr Leu Gly Thr
465 470 475 480
Thr Met Asp Thr Tyr Lys Glu Leu Ile Glu Arg Ile Pro Glu Leu Asn
485 490 495
Lys Val Ala Arg Ala Ala Ala Glu Val Ala Gly Gln Phe Val Ile Lys
500 505 510
Leu Thr His Asp Val Glu Leu Asn Leu Asp Tyr Glu Arg Tyr Asn Ser
515 520 525
Gln Leu Leu Ser Phe Val Arg Asp Leu Asn Gln Tyr Arg Ala Asp Ile
530 535 540
Lys Glu Met Gly Leu Ser Leu Gln Trp Leu Tyr Ser Ala Arg Gly Asp
545 550 555 560
Phe Phe Arg Ala Thr Ser Arg Leu Thr Thr Asp Phe Gly Asn Ala Glu
565 570 575
Lys Thr Asp Arg Phe Val Met Lys Lys Leu Asn Asp Arg Val Met Arg
580 585 590
Val Glu Tyr His Phe Leu Ser Pro Tyr Val Ser Pro Lys Glu Ser Pro
595 600 605
Phe Arg His Val Phe Trp Gly Ser Gly Ser His Thr Leu Pro Ala Leu
610 615 620
Leu Glu Asn Leu Lys Leu Arg Lys Gln Asn Asn Gly Ala Phe Asn Glu
625 630 635 640
Thr Leu Phe Arg Asn Gln Leu Ala Leu Ala Thr Trp Thr Ile Gln Gly
645 650 655
Ala Ala Asn Ala Leu Ser Gly Asp Val Trp Asp Ile Asp Asn Glu Phe
660 665 670
<210> 96
<211> 672
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of A07
<400> 96
Cys Lys Gly Val Glu Pro Lys Thr Glu Cys Glu Arg Leu Ala Gly Thr
1 5 10 15
Glu Ser Pro Val Arg Glu Glu Pro Gly Glu Asp Phe Pro Ala Ala Arg
20 25 30
Arg Leu Tyr Trp Asp Asp Leu Lys Arg Lys Leu Ser Glu Lys Leu Asp
35 40 45
Ser Thr Asp Phe Thr Gly Thr Ile Lys Leu Leu Asn Glu Asn Ser Tyr
50 55 60
Val Pro Arg Glu Ala Gly Ser Gln Lys Asp Glu Asn Leu Ala Leu Tyr
65 70 75 80
Val Glu Asn Gln Phe Arg Glu Phe Lys Leu Ser Lys Val Trp Arg Asp
85 90 95
Gln His Phe Val Lys Ile Gln Val Lys Asp Ser Ala Gln Asn Ser Val
100 105 110
Ile Ile Val Asp Lys Asn Gly Arg Leu Val Tyr Leu Val Glu Asn Pro
115 120 125
Gly Gly Tyr Val Ala Tyr Ser Lys Ala Ala Thr Val Thr Gly Lys Leu
130 135 140
Val His Ala Asn Phe Gly Thr Lys Lys Asp Phe Glu Asp Leu Tyr Thr
145 150 155 160
Pro Val Asn Gly Ser Ile Val Ile Val Arg Ala Gly Lys Ile Thr Phe
165 170 175
Ala Glu Lys Val Ala Asn Ala Glu Ser Leu Asn Ala Ile Gly Val Leu
180 185 190
Ile Tyr Met Asp Gln Thr Lys Phe Pro Ile Val Asn Ala Glu Leu Ser
195 200 205
Phe Phe Gly His Ala His Leu Gly Thr Gly Asp Pro Tyr Thr Pro Gly
210 215 220
Phe Pro Ser Phe Asn His Thr Gln Phe Pro Pro Ser Arg Ser Ser Gly
225 230 235 240
Leu Pro Asn Ile Pro Val Gln Thr Ile Ser Arg Ala Ala Ala Glu Lys
245 250 255
Leu Phe Gly Asn Met Glu Gly Asp Cys Pro Ser Asp Trp Lys Thr Asp
260 265 270
Ser Thr Cys Arg Leu Glu Thr Ser Gln Ser Lys Asn Val Lys Leu Thr
275 280 285
Val Ser Asn Val Leu Lys Glu Ile Lys Ile Leu Asn Ile Phe Gly Val
290 295 300
Ile Lys Gly Phe Val Glu Pro Asp His Tyr Val Val Val Gly Ala Gln
305 310 315 320
Arg Asp Ala Trp Gly Pro Gly Ala Ala Lys Ser Gly Val Gly Thr Ala
325 330 335
Leu Leu Leu Lys Leu Ala Gln Met Phe Ser Asp Met Val Leu Lys Asp
340 345 350
Gly Phe Gln Pro Ser Arg Ser Ile Ile Phe Ala Ser Trp Ser Ala Gly
355 360 365
Asp Phe Gly Ser Val Gly Ala Thr Glu Trp Leu Glu Gly Tyr Leu Ser
370 375 380
Ser Leu His Leu Lys Ala Phe Thr Tyr Ile Asn Leu Asp Lys Ala Val
385 390 395 400
Leu Gly Thr Ser Asn Phe Lys Val Ser Ala Ser Pro Leu Leu Tyr Thr
405 410 415
Leu Ile Glu Lys Thr Met Gln Asn Val Lys His Pro Val Thr Gly Gln
420 425 430
Phe Leu Tyr Gln Asp Ser Asn Trp Ala Ser Lys Val Glu Lys Leu Thr
435 440 445
Leu Asp Asn Ala Ala Phe Pro Phe Leu Ala Tyr Ser Gly Ile Pro Ala
450 455 460
Val Ser Phe Cys Phe Cys Glu Asp Thr Asp Tyr Pro Tyr Leu Gly Thr
465 470 475 480
Thr Met Asp Thr Tyr Lys Glu Leu Ile Glu Arg Ile Pro Glu Leu Asn
485 490 495
Lys Val Ala Arg Ala Ala Ala Glu Val Ala Gly Gln Phe Val Ile Lys
500 505 510
Leu Thr His Asp Val Glu Leu Asn Leu Asp Tyr Glu Arg Tyr Asn Ser
515 520 525
Gln Leu Leu Ser Phe Val Arg Asp Leu Asn Gln Tyr Arg Ala Asp Ile
530 535 540
Lys Glu Met Gly Leu Ser Leu Gln Trp Leu Tyr Ser Ala Arg Gly Asp
545 550 555 560
Phe Phe Arg Ala Thr Ser Arg Leu Thr Thr Asp Phe Gly Asn Ala Glu
565 570 575
Lys Thr Asp Arg Phe Val Met Lys Lys Leu Asn Asp Arg Val Met Arg
580 585 590
Val Glu Tyr His Phe Leu Ser Pro Tyr Val Ser Pro Lys Glu Ser Pro
595 600 605
Phe Arg His Val Phe Trp Gly Ser Gly Ser His Thr Leu Pro Ala Leu
610 615 620
Leu Glu Asn Leu Lys Leu Arg Lys Gln Asn Asn Gly Ala Phe Asn Glu
625 630 635 640
Thr Leu Phe Arg Asn Gln Leu Ala Leu Ala Thr Trp Thr Ile Gln Gly
645 650 655
Ala Ala Asn Ala Leu Ser Gly Asp Val Trp Asp Ile Asp Asn Glu Phe
660 665 670
<210> 97
<211> 671
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of A08
<400> 97
Cys Lys Gly Val Glu Pro Lys Thr Glu Cys Glu Arg Leu Ala Gly Thr
1 5 10 15
Glu Ser Pro Val Arg Glu Glu Pro Gly Glu Asp Phe Pro Ala Ala Arg
20 25 30
Arg Leu Tyr Trp Asp Asp Leu Lys Arg Lys Leu Ser Glu Lys Leu Asp
35 40 45
Ser Thr Asp Phe Thr Gly Thr Ile Lys Leu Leu Asn Glu Asn Ser Tyr
50 55 60
Val Pro Arg Glu Ala Gly Ser Gln Lys Asp Glu Asn Leu Ala Leu Tyr
65 70 75 80
Val Glu Asn Gln Phe Arg Glu Phe Lys Leu Ser Lys Val Trp Arg Asp
85 90 95
Gln His Phe Val Lys Ile Gln Val Lys Asp Ser Ala Gln Asn Ser Val
100 105 110
Ile Ile Val Gln Lys Asn Gly Arg Leu Asp Leu Val Glu Asn Pro Gly
115 120 125
Gly Tyr Val Ala Tyr Ser Lys Ala Ala Thr Val Thr Gly Lys Leu Val
130 135 140
His Ala Asn Phe Gly Thr Lys Lys Asp Phe Glu Asp Leu Tyr Thr Pro
145 150 155 160
Val Asn Gly Ser Ile Val Ile Val Arg Ala Gly Lys Ile Thr Phe Ala
165 170 175
Glu Lys Val Ala Asn Ala Glu Ser Leu Asn Ala Ile Gly Val Leu Ile
180 185 190
Tyr Met Asp Gln Thr Lys Phe Pro Ile Val Asn Ala Glu Leu Ser Phe
195 200 205
Phe Gly His Ala His Leu Gly Thr Gly Asp Pro Tyr Thr Pro Gly Phe
210 215 220
Pro Ser Phe Asn His Thr Gln Phe Pro Pro Ser Arg Ser Ser Gly Leu
225 230 235 240
Pro Asn Ile Pro Val Gln Thr Ile Ser Arg Ala Ala Ala Glu Lys Leu
245 250 255
Phe Gly Lys Met Glu Gly Asp Cys Pro Ser Asp Trp Lys Thr Asp Ser
260 265 270
Thr Cys Arg Met Val Thr Ser Glu Ser Gln Asn Val Lys Leu Thr Val
275 280 285
Ser Asn Val Leu Lys Glu Ile Lys Ile Leu Asn Ile Phe Gly Val Ile
290 295 300
Lys Gly Phe Val Glu Pro Asp His Tyr Val Val Val Gly Ala Gln Arg
305 310 315 320
Asp Ala Trp Gly Pro Gly Ala Ala Lys Ser Gly Val Gly Thr Ala Leu
325 330 335
Leu Leu Lys Leu Ala Gln Met Phe Ser Asp Met Val Leu Lys Asp Gly
340 345 350
Phe Gln Pro Ser Arg Ser Ile Ile Phe Ala Ser Trp Ser Ala Gly Asp
355 360 365
Phe Gly Ser Val Gly Ala Thr Glu Trp Leu Glu Gly Tyr Leu Ser Ser
370 375 380
Leu His Leu Lys Ala Phe Thr Tyr Ile Asn Leu Asp Lys Ala Val Leu
385 390 395 400
Gly Thr Ser Asn Phe Lys Val Ser Ala Ser Pro Leu Leu Tyr Thr Leu
405 410 415
Ile Glu Lys Thr Met Gln Asn Val Lys His Pro Val Thr Gly Gln Phe
420 425 430
Leu Tyr Gln Asp Ser Asn Trp Ala Ser Lys Val Glu Lys Leu Thr Leu
435 440 445
Asp Asn Ala Ala Phe Pro Phe Leu Ala Tyr Ser Gly Ile Pro Ala Val
450 455 460
Ser Phe Cys Phe Cys Glu Asp Thr Asp Tyr Pro Tyr Leu Gly Thr Thr
465 470 475 480
Met Asp Thr Tyr Lys Glu Leu Ile Glu Arg Ile Pro Glu Leu Asn Lys
485 490 495
Val Ala Arg Ala Ala Ala Glu Val Ala Gly Gln Phe Val Ile Lys Leu
500 505 510
Thr His Asp Val Glu Leu Asn Leu Asp Tyr Glu Arg Tyr Asn Ser Gln
515 520 525
Leu Leu Ser Phe Val Arg Asp Leu Asn Gln Tyr Arg Ala Asp Ile Lys
530 535 540
Glu Met Gly Leu Ser Leu Gln Trp Leu Tyr Ser Ala Arg Gly Asp Phe
545 550 555 560
Phe Arg Ala Thr Ser Arg Leu Thr Thr Asp Phe Gly Asn Ala Glu Lys
565 570 575
Thr Asp Arg Phe Val Met Lys Lys Leu Asn Asp Arg Val Met Arg Val
580 585 590
Glu Tyr His Phe Leu Ser Pro Tyr Val Ser Pro Lys Glu Ser Pro Phe
595 600 605
Arg His Val Phe Trp Gly Ser Gly Ser His Thr Leu Pro Ala Leu Leu
610 615 620
Glu Asn Leu Lys Leu Arg Lys Gln Asn Asn Gly Ala Phe Asn Glu Thr
625 630 635 640
Leu Phe Arg Asn Gln Leu Ala Leu Ala Thr Trp Thr Ile Gln Gly Ala
645 650 655
Ala Asn Ala Leu Ser Gly Asp Val Trp Asp Ile Asp Asn Glu Phe
660 665 670
<210> 98
<211> 672
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of A09
<400> 98
Cys Lys Gly Val Glu Pro Lys Thr Glu Cys Glu Arg Leu Ala Gly Thr
1 5 10 15
Glu Ser Pro Val Arg Glu Glu Pro Gly Glu Asp Phe Pro Ala Ala Arg
20 25 30
Arg Leu Tyr Trp Asp Asp Leu Lys Arg Lys Leu Ser Glu Lys Leu Asp
35 40 45
Ser Thr Asp Phe Thr Gly Thr Ile Lys Leu Leu Asn Glu Asn Ser Tyr
50 55 60
Val Pro Arg Glu Ala Gly Ser Gln Lys Asp Glu Asn Leu Ala Leu Tyr
65 70 75 80
Val Glu Asn Gln Phe Arg Glu Phe Lys Leu Ser Lys Val Trp Arg Asp
85 90 95
Gln His Phe Val Lys Ile Gln Val Lys Asp Ser Ala Gln Asn Ser Val
100 105 110
Ile Ile Val Asp Lys Asn Gly Arg Leu Val Tyr Leu Val Glu Asn Pro
115 120 125
Gly Gly Tyr Val Ala Tyr Ser Lys Pro Thr Glu Val Ser Gly Lys Leu
130 135 140
Val His Ala Asn Phe Gly Thr Lys Lys Asp Phe Glu Asp Leu Tyr Thr
145 150 155 160
Pro Val Asn Gly Ser Ile Val Ile Val Arg Ala Gly Lys Ile Thr Phe
165 170 175
Ala Glu Lys Val Ala Asn Ala Glu Ser Leu Asn Ala Ile Gly Val Leu
180 185 190
Ile Tyr Met Asp Gln Thr Lys Phe Pro Ile Val Asn Ala Glu Leu Ser
195 200 205
Phe Phe Gly His Ala His Leu Gly Thr Gly Asp Pro Tyr Thr Pro Gly
210 215 220
Phe Pro Ser Phe Asn His Thr Gln Phe Pro Pro Ser Arg Ser Ser Gly
225 230 235 240
Leu Pro Asn Ile Pro Val Gln Thr Ile Ser Arg Ala Ala Ala Glu Lys
245 250 255
Leu Phe Gly Asn Met Glu Gly Asp Cys Pro Ser Asp Trp Lys Thr Asp
260 265 270
Ser Thr Cys Arg Met Val Thr Ser Glu Ser Lys Asn Val Lys Leu Thr
275 280 285
Val Ser Asn Val Leu Lys Glu Ile Lys Ile Leu Asn Ile Phe Gly Val
290 295 300
Ile Lys Gly Phe Val Glu Pro Asp His Tyr Val Val Val Gly Ala Gln
305 310 315 320
Arg Asp Ala Trp Gly Pro Gly Ala Ala Lys Ser Gly Val Gly Thr Ala
325 330 335
Leu Leu Leu Lys Leu Ala Gln Met Phe Ser Asp Met Val Leu Lys Asp
340 345 350
Gly Phe Gln Pro Ser Arg Ser Ile Ile Phe Ala Ser Trp Ser Ala Gly
355 360 365
Asp Phe Gly Ser Val Gly Ala Thr Glu Trp Leu Glu Gly Tyr Leu Ser
370 375 380
Ser Leu His Leu Lys Ala Phe Thr Tyr Ile Asn Leu Asp Lys Ala Val
385 390 395 400
Leu Gly Thr Ser Asn Phe Lys Val Ser Ala Ser Pro Leu Leu Tyr Thr
405 410 415
Leu Ile Glu Lys Thr Met Gln Asn Val Lys His Pro Val Thr Gly Gln
420 425 430
Phe Leu Tyr Gln Asp Ser Asn Trp Ala Ser Lys Val Glu Lys Leu Thr
435 440 445
Leu Asp Asn Ala Ala Phe Pro Phe Leu Ala Tyr Ser Gly Ile Pro Ala
450 455 460
Val Ser Phe Cys Phe Cys Glu Asp Thr Asp Tyr Pro Tyr Leu Gly Thr
465 470 475 480
Thr Met Asp Thr Tyr Lys Glu Leu Ile Glu Arg Ile Pro Glu Leu Asn
485 490 495
Lys Val Ala Arg Ala Ala Ala Glu Val Ala Gly Gln Phe Val Ile Lys
500 505 510
Leu Thr His Asp Val Glu Leu Asn Leu Asp Tyr Glu Arg Tyr Asn Ser
515 520 525
Gln Leu Leu Ser Phe Val Arg Asp Leu Asn Gln Tyr Arg Ala Asp Ile
530 535 540
Lys Glu Met Gly Leu Ser Leu Gln Trp Leu Tyr Ser Ala Arg Gly Asp
545 550 555 560
Phe Phe Arg Ala Thr Ser Arg Leu Thr Thr Asp Phe Gly Asn Ala Glu
565 570 575
Lys Thr Asp Arg Phe Val Met Lys Lys Leu Asn Asp Arg Val Met Arg
580 585 590
Val Glu Tyr His Phe Leu Ser Pro Tyr Val Ser Pro Lys Glu Ser Pro
595 600 605
Phe Arg His Val Phe Trp Gly Ser Gly Ser His Thr Leu Pro Ala Leu
610 615 620
Leu Glu Asn Leu Lys Leu Arg Lys Gln Asn Asn Gly Ala Phe Asn Glu
625 630 635 640
Thr Leu Phe Arg Asn Gln Leu Ala Leu Ala Thr Trp Thr Ile Gln Gly
645 650 655
Ala Ala Asn Ala Leu Ser Gly Asp Val Trp Asp Ile Asp Asn Glu Phe
660 665 670
<210> 99
<211> 672
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of A10
<400> 99
Cys Lys Gly Val Glu Pro Lys Thr Glu Cys Glu Arg Leu Ala Gly Thr
1 5 10 15
Glu Ser Pro Val Arg Glu Glu Pro Gly Glu Asp Phe Pro Ala Ala Arg
20 25 30
Arg Leu Tyr Trp Asp Asp Leu Lys Arg Lys Leu Ser Glu Lys Leu Asp
35 40 45
Ser Thr Asp Phe Thr Gly Thr Ile Lys Leu Leu Asn Glu Asn Ser Tyr
50 55 60
Val Pro Arg Glu Ala Gly Ser Gln Lys Asp Glu Asn Leu Ala Leu Tyr
65 70 75 80
Val Glu Asn Gln Phe Arg Glu Phe Lys Leu Ser Lys Val Trp Arg Asp
85 90 95
Gln His Phe Val Lys Ile Gln Val Lys Asp Ser Ala Gln Asn Ser Val
100 105 110
Ile Ile Val Asp Lys Asn Gly Arg Leu Val Tyr Leu Val Glu Asn Pro
115 120 125
Gly Gly Tyr Val Ala Tyr Ser Lys Pro Thr Glu Val Thr Gly Lys Leu
130 135 140
Val His Ala Asn Phe Gly Thr Lys Lys Asp Phe Glu Asp Leu Tyr Thr
145 150 155 160
Pro Val Asn Gly Ser Ile Val Ile Val Arg Ala Gly Lys Ile Thr Phe
165 170 175
Ala Glu Lys Val Ala Asn Ala Glu Ser Leu Asn Ala Ile Gly Val Leu
180 185 190
Ile Tyr Met Asp Gln Thr Lys Phe Pro Ile Val Asn Ala Glu Leu Ser
195 200 205
Phe Phe Gly His Ala His Leu Gly Thr Gly Asp Pro Tyr Thr Pro Gly
210 215 220
Phe Pro Ser Phe Asn His Thr Gln Phe Pro Pro Ser Arg Ser Ser Gly
225 230 235 240
Leu Pro Asn Ile Pro Val Gln Thr Ile Ser Arg Ala Ala Ala Glu Lys
245 250 255
Leu Phe Gly Asn Met Glu Gly Asp Cys Pro Ser Asp Trp Lys Thr Asp
260 265 270
Ser Thr Cys Arg Met Val Thr Ser Glu Ser Lys Asn Val Lys Leu Thr
275 280 285
Val Ser Asn Val Leu Lys Glu Ile Lys Ile Leu Asn Ile Phe Gly Val
290 295 300
Ile Lys Gly Phe Val Glu Pro Asp His Tyr Val Val Val Gly Ala Gln
305 310 315 320
Arg Asp Ala Trp Gly Pro Gly Ala Ala Lys Ser Gly Val Gly Thr Ala
325 330 335
Leu Leu Leu Lys Leu Ala Gln Met Phe Ser Asp Met Val Leu Lys Asp
340 345 350
Gly Phe Gln Pro Ser Arg Ser Ile Ile Phe Ala Ser Trp Ser Ala Gly
355 360 365
Asp Phe Gly Ser Val Gly Ala Thr Glu Trp Leu Glu Gly Tyr Leu Ser
370 375 380
Ser Leu His Leu Lys Ala Phe Thr Tyr Ile Asn Leu Asp Lys Ala Val
385 390 395 400
Leu Gly Thr Ser Asn Phe Lys Val Ser Ala Ser Pro Leu Leu Tyr Thr
405 410 415
Leu Ile Glu Lys Thr Met Gln Asn Val Lys His Pro Val Thr Gly Gln
420 425 430
Phe Leu Tyr Gln Asp Ser Asn Trp Ala Ser Lys Val Glu Lys Leu Thr
435 440 445
Leu Asp Asn Ala Ala Phe Pro Phe Leu Ala Tyr Ser Gly Ile Pro Ala
450 455 460
Val Ser Phe Cys Phe Cys Glu Asp Thr Asp Tyr Pro Tyr Leu Gly Thr
465 470 475 480
Thr Met Asp Thr Tyr Lys Glu Leu Ile Glu Arg Ile Pro Glu Leu Asn
485 490 495
Lys Val Ala Arg Ala Ala Ala Glu Val Ala Gly Gln Phe Val Ile Lys
500 505 510
Leu Thr His Asp Val Glu Leu Asn Leu Asp Tyr Glu Arg Tyr Asn Ser
515 520 525
Gln Leu Leu Ser Phe Val Arg Asp Leu Asn Gln Tyr Arg Ala Asp Ile
530 535 540
Lys Glu Met Gly Leu Ser Leu Gln Trp Leu Tyr Ser Ala Arg Gly Asp
545 550 555 560
Phe Phe Arg Ala Thr Ser Arg Leu Thr Thr Asp Phe Gly Asn Ala Glu
565 570 575
Lys Thr Asp Arg Phe Val Met Lys Lys Leu Asn Asp Arg Val Met Arg
580 585 590
Val Glu Tyr His Phe Leu Ser Pro Tyr Val Ser Pro Lys Glu Ser Pro
595 600 605
Phe Arg His Val Phe Trp Gly Ser Gly Ser His Thr Leu Pro Ala Leu
610 615 620
Leu Glu Asn Leu Lys Leu Arg Lys Gln Asn Asn Gly Ala Phe Asn Glu
625 630 635 640
Thr Leu Phe Arg Asn Gln Leu Ala Leu Ala Thr Trp Thr Ile Gln Gly
645 650 655
Ala Ala Asn Ala Leu Ser Gly Asp Val Trp Asp Ile Asp Asn Glu Phe
660 665 670
<210> 100
<211> 673
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of A11
<400> 100
Cys Lys Gly Val Glu Pro Lys Thr Glu Cys Glu Arg Leu Ala Gly Thr
1 5 10 15
Glu Ser Pro Val Arg Glu Glu Pro Gly Glu Asp Phe Pro Ala Ala Arg
20 25 30
Arg Leu Tyr Trp Asp Asp Leu Lys Arg Lys Leu Ser Glu Lys Leu Asp
35 40 45
Ser Thr Asp Phe Thr Gly Thr Ile Lys Leu Leu Asn Glu Asn Ser Tyr
50 55 60
Val Pro Arg Glu Ala Gly Ser Gln Lys Asp Glu Asn Leu Ala Leu Tyr
65 70 75 80
Val Glu Asn Gln Phe Arg Glu Phe Lys Leu Ser Lys Val Trp Arg Asp
85 90 95
Gln His Phe Val Lys Ile Gln Val Lys Ser Ser Ile Gly Gln Asn Ser
100 105 110
Val Ile Ile Val Asp Lys Asn Gly Arg Leu Val Tyr Leu Val Glu Asn
115 120 125
Pro Gly Gly Tyr Val Ala Tyr Ser Lys Ala Ala Thr Val Thr Gly Lys
130 135 140
Leu Val His Ala Asn Phe Gly Thr Lys Lys Asp Phe Glu Asp Leu Tyr
145 150 155 160
Thr Pro Val Asn Gly Ser Ile Val Ile Val Arg Ala Gly Lys Ile Thr
165 170 175
Phe Ala Glu Lys Val Ala Asn Ala Glu Ser Leu Asn Ala Ile Gly Val
180 185 190
Leu Ile Tyr Met Asp Gln Thr Lys Phe Pro Ile Val Asn Ala Glu Leu
195 200 205
Ser Phe Phe Gly His Ala His Leu Gly Thr Gly Asp Pro Tyr Thr Pro
210 215 220
Gly Phe Pro Ser Phe Asn His Thr Gln Phe Pro Pro Ser Arg Ser Ser
225 230 235 240
Gly Leu Pro Asn Ile Pro Val Gln Thr Ile Ser Arg Ala Ala Ala Glu
245 250 255
Lys Leu Phe Gly Asn Met Glu Gly Asp Cys Pro Ser Asp Trp Lys Thr
260 265 270
Asp Ser Thr Cys Arg Met Val Thr Ser Glu Ser Lys Asn Val Lys Leu
275 280 285
Thr Val Lys Asn Val Leu Lys Glu Ile Lys Ile Leu Asn Ile Phe Gly
290 295 300
Val Ile Lys Gly Phe Val Glu Pro Asp His Tyr Val Val Val Gly Ala
305 310 315 320
Gln Arg Asp Ala Trp Gly Pro Gly Ala Ala Lys Ser Gly Val Gly Thr
325 330 335
Ala Leu Leu Leu Lys Leu Ala Gln Met Phe Ser Asp Met Val Leu Lys
340 345 350
Asp Gly Phe Gln Pro Ser Arg Ser Ile Ile Phe Ala Ser Trp Ser Ala
355 360 365
Gly Asp Phe Gly Ser Val Gly Ala Thr Glu Trp Leu Glu Gly Tyr Leu
370 375 380
Ser Ser Leu His Leu Lys Ala Phe Thr Tyr Ile Asn Leu Asp Lys Ala
385 390 395 400
Val Leu Gly Thr Ser Asn Phe Lys Val Ser Ala Ser Pro Leu Leu Tyr
405 410 415
Thr Leu Ile Glu Lys Thr Met Gln Asn Val Lys His Pro Val Thr Gly
420 425 430
Gln Phe Leu Tyr Gln Asp Ser Asn Trp Ala Ser Lys Val Glu Lys Leu
435 440 445
Thr Leu Asp Asn Ala Ala Phe Pro Phe Leu Ala Tyr Ser Gly Ile Pro
450 455 460
Ala Val Ser Phe Cys Phe Cys Glu Asp Thr Asp Tyr Pro Tyr Leu Gly
465 470 475 480
Thr Thr Met Asp Thr Tyr Lys Glu Leu Ile Glu Arg Ile Pro Glu Leu
485 490 495
Asn Lys Val Ala Arg Ala Ala Ala Glu Val Ala Gly Gln Phe Val Ile
500 505 510
Lys Leu Thr His Asp Val Glu Leu Asn Leu Asp Tyr Glu Arg Tyr Asn
515 520 525
Ser Gln Leu Leu Ser Phe Val Arg Asp Leu Asn Gln Tyr Arg Ala Asp
530 535 540
Ile Lys Glu Met Gly Leu Ser Leu Gln Trp Leu Tyr Ser Ala Arg Gly
545 550 555 560
Asp Phe Phe Arg Ala Thr Ser Arg Leu Thr Thr Asp Phe Gly Asn Ala
565 570 575
Glu Lys Thr Asp Arg Phe Val Met Lys Lys Leu Asn Asp Arg Val Met
580 585 590
Arg Val Glu Tyr His Phe Leu Ser Pro Tyr Val Ser Pro Lys Glu Ser
595 600 605
Pro Phe Arg His Val Phe Trp Gly Ser Gly Ser His Thr Leu Pro Ala
610 615 620
Leu Leu Glu Asn Leu Lys Leu Arg Lys Gln Asn Asn Gly Ala Phe Asn
625 630 635 640
Glu Thr Leu Phe Arg Asn Gln Leu Ala Leu Ala Thr Trp Thr Ile Gln
645 650 655
Gly Ala Ala Asn Ala Leu Ser Gly Asp Val Trp Asp Ile Asp Asn Glu
660 665 670
Phe
<210> 101
<211> 672
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of A12
<400> 101
Cys Lys Gly Val Glu Pro Lys Thr Glu Cys Glu Arg Leu Ala Gly Thr
1 5 10 15
Glu Ser Pro Val Arg Glu Glu Pro Gly Glu Asp Phe Pro Ala Ala Arg
20 25 30
Arg Leu Tyr Trp Asp Asp Leu Lys Arg Lys Leu Ser Glu Lys Leu Asp
35 40 45
Ser Thr Asp Phe Thr Gly Thr Ile Lys Leu Leu Asn Glu Asn Ser Tyr
50 55 60
Val Pro Arg Glu Ala Gly Ser Gln Lys Asp Glu Asn Leu Ala Leu Tyr
65 70 75 80
Val Glu Asn Gln Phe Arg Glu Phe Lys Leu Ser Lys Val Trp Arg Asp
85 90 95
Gln His Phe Val Lys Ile Gln Val Lys Asp Ser Ala Gln Asn Ser Val
100 105 110
Ile Ile Val Asp Lys Asn Gly Arg Leu Val Tyr Leu Val Glu Asn Pro
115 120 125
Glu Gly Tyr Val Ala Tyr Ser Lys Ala Ala Thr Val Thr Gly Lys Leu
130 135 140
Val His Ala Asn Phe Gly Thr Lys Lys Asp Phe Glu Asp Leu Tyr Thr
145 150 155 160
Pro Val Asn Gly Ser Ile Val Ile Val Arg Ala Gly Lys Ile Thr Phe
165 170 175
Ala Glu Lys Val Ala Asn Ala Glu Ser Leu Asn Ala Ile Gly Val Leu
180 185 190
Ile Tyr Met Asp Gln Thr Lys Phe Pro Ile Val Asn Ala Glu Leu Ala
195 200 205
Phe Phe Gly His Ala His Leu Gly Thr Gly Asp Pro Tyr Thr Pro Gly
210 215 220
Phe Pro Ser Phe Asn His Thr Gln Phe Pro Pro Ser Arg Ser Ser Gly
225 230 235 240
Leu Pro Asn Ile Pro Val Gln Thr Ile Ser Arg Ala Ala Ala Glu Lys
245 250 255
Leu Phe Gly Asn Met Glu Gly Asp Cys Pro Ser Asp Trp Lys Thr Asp
260 265 270
Ser Thr Cys Arg Met Val Thr Ser Glu Ser Lys Asn Val Lys Leu Thr
275 280 285
Val Ser Asn Val Leu Lys Glu Ile Lys Ile Leu Asn Ile Phe Gly Val
290 295 300
Ile Lys Gly Phe Val Glu Pro Asp His Tyr Val Val Val Gly Ala Gln
305 310 315 320
Arg Asp Ala Trp Gly Pro Gly Ala Ala Lys Ser Gly Val Gly Thr Ala
325 330 335
Leu Leu Leu Lys Leu Ala Gln Met Phe Ser Asp Met Val Leu Lys Asp
340 345 350
Gly Phe Gln Pro Ser Arg Ser Ile Ile Phe Ala Ser Trp Ser Ala Gly
355 360 365
Asp Phe Gly Ser Val Gly Ala Thr Glu Trp Leu Glu Gly Tyr Leu Ser
370 375 380
Ser Leu His Leu Lys Ala Phe Thr Tyr Ile Asn Leu Asp Lys Ala Val
385 390 395 400
Leu Gly Thr Ser Asn Phe Lys Val Ser Ala Ser Pro Leu Leu Tyr Thr
405 410 415
Leu Ile Glu Lys Thr Met Gln Asn Val Lys His Pro Val Thr Gly Gln
420 425 430
Phe Leu Tyr Gln Asp Ser Asn Trp Ala Ser Lys Val Glu Lys Leu Thr
435 440 445
Leu Asp Asn Ala Ala Phe Pro Phe Leu Ala Tyr Ser Gly Ile Pro Ala
450 455 460
Val Ser Phe Cys Phe Cys Glu Asp Thr Asp Tyr Pro Tyr Leu Gly Thr
465 470 475 480
Thr Met Asp Thr Tyr Lys Glu Leu Ile Glu Arg Ile Pro Glu Leu Asn
485 490 495
Lys Val Ala Arg Ala Ala Ala Glu Val Ala Gly Gln Phe Val Ile Lys
500 505 510
Leu Thr His Asp Val Glu Leu Asn Leu Asp Tyr Glu Arg Tyr Asn Ser
515 520 525
Gln Leu Leu Ser Phe Val Arg Asp Leu Asn Gln Tyr Arg Ala Asp Ile
530 535 540
Lys Glu Met Gly Leu Ser Leu Gln Trp Leu Tyr Ser Ala Arg Gly Asp
545 550 555 560
Phe Phe Arg Ala Thr Ser Arg Leu Thr Thr Asp Phe Gly Asn Ala Glu
565 570 575
Lys Thr Asp Arg Phe Val Met Lys Lys Leu Asn Asp Arg Val Met Arg
580 585 590
Val Glu Tyr His Phe Leu Ser Pro Tyr Val Ser Pro Lys Glu Ser Pro
595 600 605
Phe Arg His Val Phe Trp Gly Ser Gly Ser His Thr Leu Pro Ala Leu
610 615 620
Leu Glu Asn Leu Lys Leu Arg Lys Gln Asn Asn Gly Ala Phe Asn Glu
625 630 635 640
Thr Leu Phe Arg Asn Gln Leu Ala Leu Ala Thr Trp Thr Ile Gln Gly
645 650 655
Ala Ala Asn Ala Leu Ser Gly Asp Val Trp Asp Ile Asp Asn Glu Phe
660 665 670
<210> 102
<211> 672
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of A13
<400> 102
Cys Lys Gly Val Glu Pro Lys Thr Glu Cys Glu Arg Leu Ala Gly Thr
1 5 10 15
Glu Ser Pro Val Arg Glu Glu Pro Gly Glu Asp Phe Pro Ala Ala Arg
20 25 30
Arg Leu Tyr Trp Asp Asp Leu Lys Arg Lys Leu Ser Glu Lys Leu Asp
35 40 45
Ser Thr Asp Phe Thr Gly Thr Ile Lys Leu Leu Asn Glu Asn Ser Tyr
50 55 60
Val Pro Arg Glu Ala Gly Ser Gln Lys Asp Glu Asn Leu Ala Leu Tyr
65 70 75 80
Val Glu Asn Gln Phe Arg Glu Phe Lys Leu Ser Lys Val Trp Arg Asp
85 90 95
Gln His Phe Val Lys Ile Gln Val Lys Asp Ser Ala Gln Asn Ser Val
100 105 110
Ile Ile Val Gln Ser Asn Gly Arg Leu Val Tyr Leu Val Glu Asn Pro
115 120 125
Gly Gly Tyr Val Ala Tyr Ser Lys Ala Ala Thr Val Thr Gly Lys Leu
130 135 140
Val His Ala Asn Phe Gly Thr Lys Lys Asp Phe Glu Asp Leu Tyr Thr
145 150 155 160
Pro Val Asn Gly Ser Ile Val Ile Val Arg Ala Gly Lys Ile Thr Phe
165 170 175
Ala Glu Lys Val Ala Asn Ala Glu Ser Leu Asn Ala Ile Gly Val Leu
180 185 190
Ile Tyr Met Asp Gln Thr Lys Phe Pro Ile Val Asn Ala Glu Leu Ser
195 200 205
Phe Phe Gly His Ala His Leu Gly Thr Gly Asp Pro Tyr Thr Pro Gly
210 215 220
Phe Pro Ser Phe Asn His Thr Gln Phe Pro Pro Ser Arg Ser Ser Gly
225 230 235 240
Leu Pro Asn Ile Pro Val Gln Thr Ile Ser Arg Ala Ala Ala Glu Lys
245 250 255
Leu Phe Gly Asn Met Glu Gly Asp Cys Pro Ser Asp Trp Lys Thr Asp
260 265 270
Ser Thr Cys Arg Met Val Thr Ser Glu Ser Lys Asn Val Lys Leu Thr
275 280 285
Val Ser Asn Val Leu Lys Glu Ile Lys Ile Leu Asn Ile Phe Gly Val
290 295 300
Ile Lys Gly Phe Val Glu Pro Asp His Tyr Val Val Val Gly Ala Gln
305 310 315 320
Arg Asp Ala Trp Gly Pro Gly Ala Ala Lys Ser Gly Val Gly Thr Ala
325 330 335
Leu Leu Leu Lys Leu Ala Gln Met Phe Ser Asp Met Val Leu Lys Asp
340 345 350
Gly Phe Gln Pro Ser Arg Ser Ile Ile Phe Ala Ser Trp Ser Ala Gly
355 360 365
Asp Phe Gly Ser Val Gly Ala Thr Glu Trp Leu Glu Gly Tyr Leu Ser
370 375 380
Ser Leu His Leu Lys Ala Phe Thr Tyr Ile Asn Leu Asp Lys Ala Val
385 390 395 400
Leu Gly Thr Ser Asn Phe Lys Val Ser Ala Ser Pro Leu Leu Tyr Thr
405 410 415
Leu Ile Glu Lys Thr Met Gln Asn Val Lys His Pro Val Thr Gly Gln
420 425 430
Phe Leu Tyr Gln Asp Ser Asn Trp Ala Ser Lys Val Glu Lys Leu Thr
435 440 445
Leu Asp Asn Ala Ala Phe Pro Phe Leu Ala Tyr Ser Gly Ile Pro Ala
450 455 460
Val Ser Phe Cys Phe Cys Glu Asp Thr Asp Tyr Pro Tyr Leu Gly Thr
465 470 475 480
Thr Met Asp Thr Tyr Lys Glu Leu Ile Glu Arg Ile Pro Glu Leu Asn
485 490 495
Lys Val Ala Arg Ala Ala Ala Glu Val Ala Gly Gln Phe Val Ile Lys
500 505 510
Leu Thr His Asp Val Glu Leu Asn Leu Asp Tyr Glu Arg Tyr Asn Ser
515 520 525
Gln Leu Leu Ser Phe Val Arg Asp Leu Asn Gln Tyr Arg Ala Asp Ile
530 535 540
Lys Glu Met Gly Leu Ser Leu Gln Trp Leu Tyr Ser Ala Arg Gly Asp
545 550 555 560
Phe Phe Arg Ala Thr Ser Arg Leu Thr Thr Asp Phe Gly Asn Ala Glu
565 570 575
Lys Thr Asp Arg Phe Val Met Lys Lys Leu Asn Asp Arg Val Met Arg
580 585 590
Val Glu Tyr His Phe Leu Ser Pro Tyr Val Ser Pro Lys Glu Ser Pro
595 600 605
Phe Arg His Val Phe Trp Gly Ser Gly Ser His Thr Leu Pro Ala Leu
610 615 620
Leu Glu Asn Leu Lys Leu Arg Lys Gln Asn Asn Gly Ala Phe Asn Glu
625 630 635 640
Thr Leu Phe Arg Asn Gln Leu Ala Leu Ala Thr Trp Thr Ile Gln Gly
645 650 655
Ala Ala Asn Ala Leu Ser Gly Asp Val Trp Asp Ile Asp Asn Glu Phe
660 665 670
<210> 103
<211> 672
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of A14
<400> 103
Cys Lys Gly Val Glu Pro Lys Thr Glu Cys Glu Arg Leu Ala Gly Thr
1 5 10 15
Glu Ser Pro Val Arg Glu Glu Pro Gly Glu Asp Phe Pro Ala Ala Arg
20 25 30
Arg Leu Tyr Trp Asp Asp Leu Lys Arg Lys Leu Ser Glu Lys Leu Asp
35 40 45
Ser Thr Asp Phe Thr Gly Thr Ile Lys Leu Leu Asn Glu Asn Ser Tyr
50 55 60
Val Pro Arg Glu Ala Gly Ser Gln Lys Asp Glu Asn Leu Ala Leu Tyr
65 70 75 80
Val Glu Asn Gln Phe Arg Glu Phe Lys Leu Ser Lys Val Trp Arg Asp
85 90 95
Gln His Phe Val Lys Ile Gln Val Lys Asp Ser Ala Gln Asn Ser Val
100 105 110
Ile Ile Val Gln Ser Asn Gly Arg Leu Val Tyr Leu Val Glu Asn Pro
115 120 125
Gly Gly Tyr Val Ala Tyr Ser Lys Ala Ala Thr Val Thr Gly Lys Leu
130 135 140
Val His Ala Asn Phe Gly Thr Lys Lys Asp Phe Glu Asp Leu Tyr Thr
145 150 155 160
Pro Val Asn Gly Ser Ile Val Ile Val Arg Ala Gly Lys Ile Thr Phe
165 170 175
Ala Glu Lys Val Ala Asn Ala Glu Ser Leu Asn Ala Ile Gly Val Leu
180 185 190
Ile Tyr Met Asp Gln Thr Lys Phe Pro Ile Val Asn Ala Glu Leu Ser
195 200 205
Phe Phe Gly His Ala His Leu Gly Thr Gly Asp Pro Tyr Thr Pro Gly
210 215 220
Phe Pro Ser Phe Asn His Thr Gln Phe Pro Pro Ser Arg Ser Ser Gly
225 230 235 240
Leu Pro Asn Ile Pro Val Gln Thr Ile Ser Arg Ala Ala Ala Glu Lys
245 250 255
Leu Phe Gly Asn Met Glu Gly Asp Cys Pro Ser Asp Trp Lys Thr Asp
260 265 270
Ser Thr Cys Arg Met Val Thr Ser Gln Ser Lys Asn Val Lys Leu Thr
275 280 285
Val Ser Asn Val Leu Lys Glu Ile Lys Ile Leu Asn Ile Phe Gly Val
290 295 300
Ile Lys Gly Phe Val Glu Pro Asp His Tyr Val Val Val Gly Ala Gln
305 310 315 320
Arg Asp Ala Trp Gly Pro Gly Ala Ala Lys Ser Gly Val Gly Thr Ala
325 330 335
Leu Leu Leu Lys Leu Ala Gln Met Phe Ser Asp Met Val Leu Lys Asp
340 345 350
Gly Phe Gln Pro Ser Arg Ser Ile Ile Phe Ala Ser Trp Ser Ala Gly
355 360 365
Asp Phe Gly Ser Val Gly Ala Thr Glu Trp Leu Glu Gly Tyr Leu Ser
370 375 380
Ser Leu His Leu Lys Ala Phe Thr Tyr Ile Asn Leu Asp Lys Ala Val
385 390 395 400
Leu Gly Thr Ser Asn Phe Lys Val Ser Ala Ser Pro Leu Leu Tyr Thr
405 410 415
Leu Ile Glu Lys Thr Met Gln Asn Val Lys His Pro Val Thr Gly Gln
420 425 430
Phe Leu Tyr Gln Asp Ser Asn Trp Ala Ser Lys Val Glu Lys Leu Thr
435 440 445
Leu Asp Asn Ala Ala Phe Pro Phe Leu Ala Tyr Ser Gly Ile Pro Ala
450 455 460
Val Ser Phe Cys Phe Cys Glu Asp Thr Asp Tyr Pro Tyr Leu Gly Thr
465 470 475 480
Thr Met Asp Thr Tyr Lys Glu Leu Ile Glu Arg Ile Pro Glu Leu Asn
485 490 495
Lys Val Ala Arg Ala Ala Ala Glu Val Ala Gly Gln Phe Val Ile Lys
500 505 510
Leu Thr His Asp Val Glu Leu Asn Leu Asp Tyr Glu Arg Tyr Asn Ser
515 520 525
Gln Leu Leu Ser Phe Val Arg Asp Leu Asn Gln Tyr Arg Ala Asp Ile
530 535 540
Lys Glu Met Gly Leu Ser Leu Gln Trp Leu Tyr Ser Ala Arg Gly Asp
545 550 555 560
Phe Phe Arg Ala Thr Ser Arg Leu Thr Thr Asp Phe Gly Asn Ala Glu
565 570 575
Lys Thr Asp Arg Phe Val Met Lys Lys Leu Asn Asp Arg Val Met Arg
580 585 590
Val Glu Tyr His Phe Leu Ser Pro Tyr Val Ser Pro Lys Glu Ser Pro
595 600 605
Phe Arg His Val Phe Trp Gly Ser Gly Ser His Thr Leu Pro Ala Leu
610 615 620
Leu Glu Asn Leu Lys Leu Arg Lys Gln Asn Asn Gly Ala Phe Asn Glu
625 630 635 640
Thr Leu Phe Arg Asn Gln Leu Ala Leu Ala Thr Trp Thr Ile Gln Gly
645 650 655
Ala Ala Asn Ala Leu Ser Gly Asp Val Trp Asp Ile Asp Asn Glu Phe
660 665 670
<210> 104
<211> 672
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of A15
<400> 104
Cys Lys Gly Val Glu Pro Lys Thr Glu Cys Glu Arg Leu Ala Gly Thr
1 5 10 15
Glu Ser Pro Val Arg Glu Glu Pro Gly Glu Asp Phe Pro Ala Ala Arg
20 25 30
Arg Leu Tyr Trp Asp Asp Leu Lys Arg Lys Leu Ser Glu Lys Leu Asp
35 40 45
Ser Thr Asp Phe Thr Gly Thr Ile Lys Leu Leu Asn Glu Asn Ser Tyr
50 55 60
Val Pro Arg Glu Ala Gly Ser Gln Lys Asp Glu Asn Leu Ala Leu Tyr
65 70 75 80
Val Glu Asn Gln Phe Arg Glu Phe Lys Leu Ser Lys Val Trp Arg Asp
85 90 95
Gln His Phe Val Lys Ile Gln Val Lys Asp Ser Ala Gln Asn Met Val
100 105 110
Thr Ile Val Asp Lys Asn Gly Arg Leu Val Tyr Pro Val Glu Ser Pro
115 120 125
Gly Gly Tyr Val Ala Tyr Ser Lys Ala Ala Thr Val Thr Gly Lys Leu
130 135 140
Val His Ala Asn Phe Gly Thr Lys Lys Asp Phe Glu Asp Leu Tyr Thr
145 150 155 160
Pro Val Asn Gly Ser Ile Val Ile Val Arg Ala Gly Lys Ile Thr Phe
165 170 175
Ala Glu Lys Val Ala Asn Ala Glu Ser Leu Asn Ala Ile Gly Val Leu
180 185 190
Ile Tyr Met Asp Gln Thr Lys Phe Pro Ile Val Asn Ala Glu Leu Ser
195 200 205
Phe Phe Gly His Ala His Leu Gly Thr Gly Asp Pro Tyr Thr Pro Gly
210 215 220
Phe Pro Ser Phe Asn His Thr Gln Phe Pro Pro Ser Arg Ser Ser Gly
225 230 235 240
Leu Pro Asn Ile Pro Val Gln Thr Ile Ser Arg Ala Ala Ala Glu Lys
245 250 255
Leu Phe Gly Asn Met Glu Gly Asp Cys Pro Ser Asp Trp Lys Thr Asp
260 265 270
Ser Thr Cys Arg Met Val Thr Ser Glu Ser Lys Asn Val Lys Leu Thr
275 280 285
Val Ser Asn Val Leu Lys Glu Ile Lys Ile Leu Asn Ile Phe Gly Val
290 295 300
Ile Lys Gly Phe Val Glu Pro Asp His Tyr Val Val Val Gly Ala Gln
305 310 315 320
Arg Asp Ala Trp Gly Pro Gly Ala Ala Lys Ser Gly Val Gly Thr Ala
325 330 335
Leu Leu Leu Lys Leu Ala Gln Met Phe Ser Asp Met Val Leu Lys Asp
340 345 350
Gly Phe Gln Pro Ser Arg Ser Ile Ile Phe Ala Ser Trp Ser Ala Gly
355 360 365
Asp Phe Gly Ser Val Gly Ala Thr Glu Trp Leu Glu Gly Tyr Leu Ser
370 375 380
Ser Leu His Leu Lys Ala Phe Thr Tyr Ile Asn Leu Asp Lys Ala Val
385 390 395 400
Leu Gly Thr Ser Asn Phe Lys Val Ser Ala Ser Pro Leu Leu Tyr Thr
405 410 415
Leu Ile Glu Lys Thr Met Gln Asn Val Lys His Pro Val Thr Gly Gln
420 425 430
Phe Leu Tyr Gln Asp Ser Asn Trp Ala Ser Lys Val Glu Lys Leu Thr
435 440 445
Leu Asp Asn Ala Ala Phe Pro Phe Leu Ala Tyr Ser Gly Ile Pro Ala
450 455 460
Val Ser Phe Cys Phe Cys Glu Asp Thr Asp Tyr Pro Tyr Leu Gly Thr
465 470 475 480
Thr Met Asp Thr Tyr Lys Glu Leu Ile Glu Arg Ile Pro Glu Leu Asn
485 490 495
Lys Val Ala Arg Ala Ala Ala Glu Val Ala Gly Gln Phe Val Ile Lys
500 505 510
Leu Thr His Asp Val Glu Leu Asn Leu Asp Tyr Glu Arg Tyr Asn Ser
515 520 525
Gln Leu Leu Ser Phe Val Arg Asp Leu Asn Gln Tyr Arg Ala Asp Ile
530 535 540
Lys Glu Met Gly Leu Ser Leu Gln Trp Leu Tyr Ser Ala Arg Gly Asp
545 550 555 560
Phe Phe Arg Ala Thr Ser Arg Leu Thr Thr Asp Phe Gly Asn Ala Glu
565 570 575
Lys Thr Asp Arg Phe Val Met Lys Lys Leu Asn Asp Arg Val Met Arg
580 585 590
Val Glu Tyr His Phe Leu Ser Pro Tyr Val Ser Pro Lys Glu Ser Pro
595 600 605
Phe Arg His Val Phe Trp Gly Ser Gly Ser His Thr Leu Pro Ala Leu
610 615 620
Leu Glu Asn Leu Lys Leu Arg Lys Gln Asn Asn Gly Ala Phe Asn Glu
625 630 635 640
Thr Leu Phe Arg Asn Gln Leu Ala Leu Ala Thr Trp Thr Ile Gln Gly
645 650 655
Ala Ala Asn Ala Leu Ser Gly Asp Val Trp Asp Ile Asp Asn Glu Phe
660 665 670
<210> 105
<211> 672
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of A16
<400> 105
Cys Lys Gly Val Glu Pro Lys Thr Glu Cys Glu Arg Leu Ala Gly Thr
1 5 10 15
Glu Ser Pro Val Arg Glu Glu Pro Gly Glu Asp Phe Pro Ala Ala Arg
20 25 30
Arg Leu Tyr Trp Asp Asp Leu Lys Arg Lys Leu Ser Glu Lys Leu Asp
35 40 45
Ser Thr Asp Phe Thr Gly Thr Ile Lys Leu Leu Asn Glu Asn Ser Tyr
50 55 60
Val Pro Arg Glu Ala Gly Ser Gln Lys Asp Glu Asn Leu Ala Leu Tyr
65 70 75 80
Val Glu Asn Gln Phe Arg Glu Phe Lys Leu Ser Lys Val Trp Arg Asp
85 90 95
Gln His Phe Val Lys Ile Gln Val Lys Asp Ser Ala Gln Asn Met Val
100 105 110
Ile Ile Val Asp Lys Asn Gly Arg Leu Val Tyr Leu Val Glu Asn Pro
115 120 125
Gly Gly Tyr Val Ala Tyr Ser Lys Ala Ala Glu Val Thr Gly Lys Leu
130 135 140
Val His Ala Asn Phe Gly Thr Lys Lys Asp Phe Glu Asp Leu Tyr Thr
145 150 155 160
Pro Val Asn Gly Ser Ile Val Ile Val Arg Ala Gly Lys Ile Thr Phe
165 170 175
Ala Glu Lys Val Ala Asn Ala Glu Ser Leu Asn Ala Ile Gly Val Leu
180 185 190
Ile Tyr Met Asp Gln Thr Lys Phe Pro Ile Val Asn Ala Glu Leu Ser
195 200 205
Phe Phe Gly His Ala His Leu Gly Thr Gly Asp Pro Tyr Thr Pro Gly
210 215 220
Phe Pro Ser Phe Asn His Thr Gln Phe Pro Pro Ser Arg Ser Ser Gly
225 230 235 240
Leu Pro Asn Ile Pro Val Gln Thr Ile Ser Arg Ala Ala Ala Glu Lys
245 250 255
Leu Phe Gly Asn Met Glu Gly Asp Cys Pro Ser Asp Trp Lys Thr Asp
260 265 270
Ser Thr Cys Arg Met Val Thr Ser Glu Ser Lys Asn Val Lys Leu Ile
275 280 285
Val Ser Asn Val Leu Lys Glu Ile Lys Ile Leu Asn Ile Phe Gly Val
290 295 300
Ile Lys Gly Phe Val Glu Pro Asp His Tyr Val Val Val Gly Ala Gln
305 310 315 320
Arg Asp Ala Trp Gly Pro Gly Ala Ala Lys Ser Gly Val Gly Thr Ala
325 330 335
Leu Leu Leu Lys Leu Ala Gln Met Phe Ser Asp Met Val Leu Lys Asp
340 345 350
Gly Phe Gln Pro Ser Arg Ser Ile Ile Phe Ala Ser Trp Ser Ala Gly
355 360 365
Asp Phe Gly Ser Val Gly Ala Thr Glu Trp Leu Glu Gly Tyr Leu Ser
370 375 380
Ser Leu His Leu Lys Ala Phe Thr Tyr Ile Asn Leu Asp Lys Ala Val
385 390 395 400
Leu Gly Thr Ser Asn Phe Lys Val Ser Ala Ser Pro Leu Leu Tyr Thr
405 410 415
Leu Ile Glu Lys Thr Met Gln Asn Val Lys His Pro Val Thr Gly Gln
420 425 430
Phe Leu Tyr Gln Asp Ser Asn Trp Ala Ser Lys Val Glu Lys Leu Thr
435 440 445
Leu Asp Asn Ala Ala Phe Pro Phe Leu Ala Tyr Ser Gly Ile Pro Ala
450 455 460
Val Ser Phe Cys Phe Cys Glu Asp Thr Asp Tyr Pro Tyr Leu Gly Thr
465 470 475 480
Thr Met Asp Thr Tyr Lys Glu Leu Ile Glu Arg Ile Pro Glu Leu Asn
485 490 495
Lys Val Ala Arg Ala Ala Ala Glu Val Ala Gly Gln Phe Val Ile Lys
500 505 510
Leu Thr His Asp Val Glu Leu Asn Leu Asp Tyr Glu Arg Tyr Asn Ser
515 520 525
Gln Leu Leu Ser Phe Val Arg Asp Leu Asn Gln Tyr Arg Ala Asp Ile
530 535 540
Lys Glu Met Gly Leu Ser Leu Gln Trp Leu Tyr Ser Ala Arg Gly Asp
545 550 555 560
Phe Phe Arg Ala Thr Ser Arg Leu Thr Thr Asp Phe Gly Asn Ala Glu
565 570 575
Lys Thr Asp Arg Phe Val Met Lys Lys Leu Asn Asp Arg Val Met Arg
580 585 590
Val Glu Tyr His Phe Leu Ser Pro Tyr Val Ser Pro Lys Glu Ser Pro
595 600 605
Phe Arg His Val Phe Trp Gly Ser Gly Ser His Thr Leu Pro Ala Leu
610 615 620
Leu Glu Asn Leu Lys Leu Arg Lys Gln Asn Asn Gly Ala Phe Asn Glu
625 630 635 640
Thr Leu Phe Arg Asn Gln Leu Ala Leu Ala Thr Trp Thr Ile Gln Gly
645 650 655
Ala Ala Asn Ala Leu Ser Gly Asp Val Trp Asp Ile Asp Asn Glu Phe
660 665 670
<210> 106
<211> 122
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of VH starting with TfR1071 EVQL
<400> 106
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser Phe Asp Lys Tyr
20 25 30
Thr Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Val Ile Ser Asn Gly Gly Val Ser Thr Asp Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Val Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Met Ala Thr Tyr Tyr Cys
85 90 95
Ala Arg Ala Ser Gln Pro Trp Leu Tyr Arg Thr Gly Ala Asp Val Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 107
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of TfR1007 HCDR1
<400> 107
Gly Tyr Ser Met Ser
1 5
<210> 108
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of TfR1007 HCDR2
<400> 108
Ser Leu Ser Asn Gly Gly Gly Ser Ser Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 109
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of TfR1007 HCDR3
<400> 109
Thr Leu Gly Ala Tyr Tyr Ile Lys Ser Ala Met Asp Val
1 5 10
<210> 110
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of Cetuximab VH
<400> 110
Gln Val Gln Leu Lys Gln Ser Gly Pro Gly Leu Val Gln Pro Ser Gln
1 5 10 15
Ser Leu Ser Ile Thr Cys Thr Val Ser Gly Phe Ser Leu Thr Asn Tyr
20 25 30
Gly Val His Trp Val Arg Gln Ser Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Val Ile Trp Ser Gly Gly Asn Thr Asp Tyr Asn Thr Pro Phe Thr
50 55 60
Ser Arg Leu Ser Ile Asn Lys Asp Asn Ser Lys Ser Gln Val Phe Phe
65 70 75 80
Lys Met Asn Ser Leu Gln Ser Asn Asp Thr Ala Ile Tyr Tyr Cys Ala
85 90 95
Arg Ala Leu Thr Tyr Tyr Asp Tyr Glu Phe Ala Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ala
115
<210> 111
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of Cetuximab VL
<400> 111
Asp Ile Leu Leu Thr Gln Ser Pro Val Ile Leu Ser Val Ser Pro Gly
1 5 10 15
Glu Arg Val Ser Phe Ser Cys Arg Ala Ser Gln Ser Ile Gly Thr Asn
20 25 30
Ile His Trp Tyr Gln Gln Arg Thr Asn Gly Ser Pro Arg Leu Leu Ile
35 40 45
Lys Tyr Ala Ser Glu Ser Ile Ser Gly Ile Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Ser Ile Asn Ser Val Glu Ser
65 70 75 80
Glu Asp Ile Ala Asp Tyr Tyr Cys Gln Gln Asn Asn Asn Trp Pro Thr
85 90 95
Thr Phe Gly Ala Gly Thr Lys Leu Glu Leu Lys
100 105
<210> 112
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of Panitumumab VH
<400> 112
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Val Ser Ser Gly
20 25 30
Asp Tyr Tyr Trp Thr Trp Ile Arg Gln Ser Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly His Ile Tyr Tyr Ser Gly Asn Thr Asn Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Leu Thr Ile Ser Ile Asp Thr Ser Lys Thr Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Ile Tyr Tyr
85 90 95
Cys Val Arg Asp Arg Val Thr Gly Ala Phe Asp Ile Trp Gly Gln Gly
100 105 110
Thr Met Val Thr Val Ser Ser
115
<210> 113
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of Panitumumab VL
<400> 113
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asp Ile Ser Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Phe Cys Gln His Phe Asp His Leu Pro Leu
85 90 95
Ala Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 114
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of Necitumumab VH
<400> 114
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Gly
20 25 30
Asp Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Ile Tyr Tyr Ser Gly Ser Thr Asp Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Met Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Val Asn Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Val Ser Ile Phe Gly Val Gly Thr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 115
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of Necitumumab VL
<400> 115
Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Arg Ala Thr Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys His Gln Tyr Gly Ser Thr Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Ala Glu Ile Lys
100 105
<210> 116
<211> 123
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of Nimotuzumab VH
<400> 116
Gln Val Gln Leu Gln Gln Pro Gly Ala Glu Leu Val Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Leu Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asn Tyr
20 25 30
Tyr Ile Tyr Trp Val Lys Gln Arg Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Gly Ile Asn Pro Thr Ser Gly Gly Ser Asn Phe Asn Glu Lys Phe
50 55 60
Lys Thr Lys Ala Thr Leu Thr Val Asp Glu Ser Ser Thr Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Tyr Cys
85 90 95
Thr Arg Gln Gly Leu Trp Phe Asp Ser Asp Gly Arg Gly Phe Asp Phe
100 105 110
Trp Gly Gln Gly Thr Thr Leu Thr Val Ser Ser
115 120
<210> 117
<211> 112
<212> PRT
<213> Artificial Sequence
<220>
<223> description of the artificial sequence: the amino acid
sequence
of Nimotuzumab VL
<400> 117
Asp Val Leu Met Thr Gln Ile Pro Leu Ser Leu Pro Val Ser Leu Gly
1 5 10 15
Asp Gln Ala Ser Ile Ser Cys Arg Ser Ser Gln Asn Ile Val His Ser
20 25 30
Asn Gly Asn Thr Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Asn Leu Leu Ile Tyr Lys Val Ser Asn Arg Glu Ser Gly Val Pro
50 55 60
Asp Arg Phe Arg Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Leu Gly Val Tyr Tyr Cys Phe Gln Tyr
85 90 95
Ser His Val Pro Trp Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105 110
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| БИСПЕЦИФИЧЕСКОЕ АНТИТЕЛО АНТИ-EGFR/АНТИ-4-1ВВ И ЕГО ПРИМЕНЕНИЕ | 2020 |
|
RU2831836C2 |
| Биспецифическое антитело анти-HER2/анти-4-1BB и его применение | 2020 |
|
RU2831838C2 |
| БИФУНКЦИОНАЛЬНЫЕ БЕЛКИ, КОМБИНИРУЮЩИЕ БЛОКАДУ КОНТРОЛЬНОЙ ТОЧКИ, ДЛЯ ТАРГЕТНОЙ ТЕРАПИИ | 2019 |
|
RU2756899C1 |
| ИНДУЦИРУЮЩИЙ ЦИТОТОКСИЧНОСТЬ ТЕРАПЕВТИЧЕСКИЙ АГЕНТ | 2022 |
|
RU2824285C2 |
| МОНОСПЕЦИФИЧЕСКИЕ И БИСПЕЦИФИЧЕСКИЕ БЕЛКИ С РЕГУЛИРОВАНИЕМ ИММУННОЙ КОНТРОЛЬНОЙ ТОЧКИ ДЛЯ ЛЕЧЕНИЯ РАКА | 2018 |
|
RU2793167C2 |
| НАЦЕЛЕННЫЕ НА ОПУХОЛЬ АГОНИСТИЧЕСКИЕ CD28-АНТИГЕНСВЯЗЫВАЮЩИЕ МОЛЕКУЛЫ | 2019 |
|
RU2837821C1 |
| БИСПЕЦИФИЧЕСКОЕ АНТИТЕЛО ПРОТИВ ВИРУСА БЕШЕНСТВА И ЕГО ПРИМЕНЕНИЕ | 2019 |
|
RU2764740C1 |
| НАЦЕЛЕННЫЕ НА ОПУХОЛЬ АГОНИСТИЧЕСКИЕ CD28-АНТИГЕНСВЯЗЫВАЮЩИЕ МОЛЕКУЛЫ | 2019 |
|
RU2808030C2 |
| БИСПЕЦИФИЧЕСКИЕ АНТИТЕЛА К ЧЕЛОВЕЧЕСКОМУ БЕТА-АМИЛОИДУ/ЧЕЛОВЕЧЕСКОМУ РЕЦЕПТОРУ ТРАНСФЕРРИНА И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2016 |
|
RU2730682C1 |
| СПОСОБ ПОЛУЧЕНИЯ ПОЛИПЕПТИДНОГО ГЕТЕРОМУЛЬТИМЕРА | 2014 |
|
RU2730594C2 |
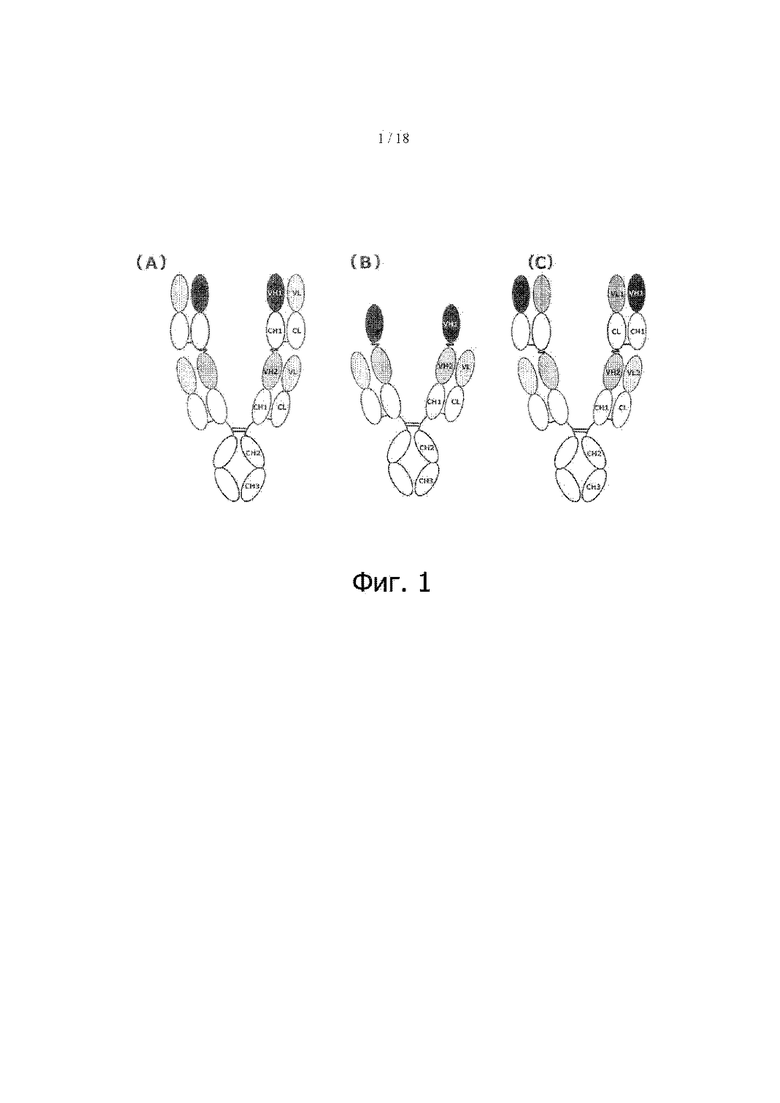
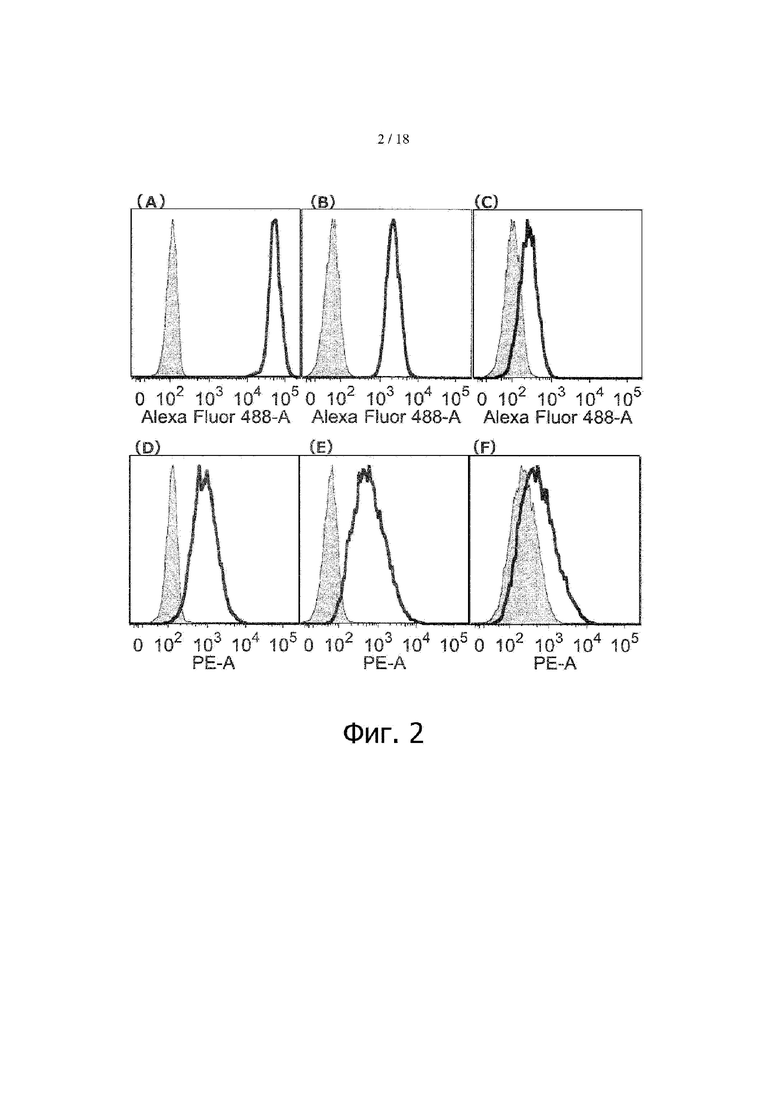
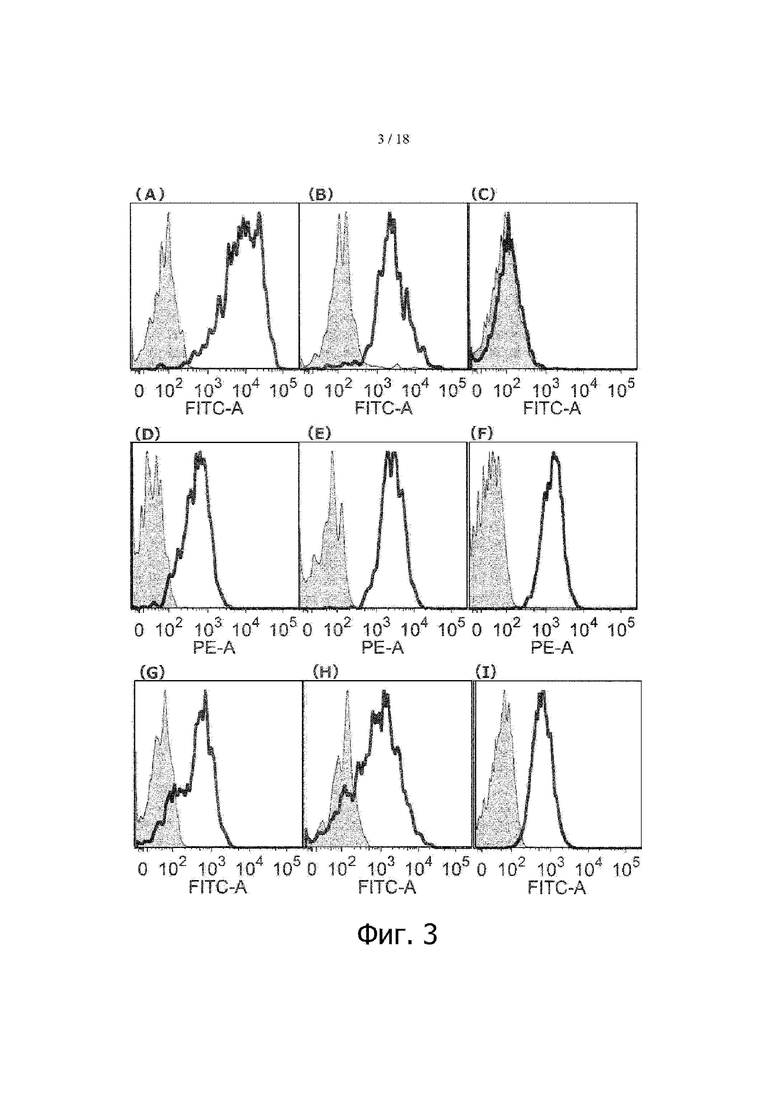
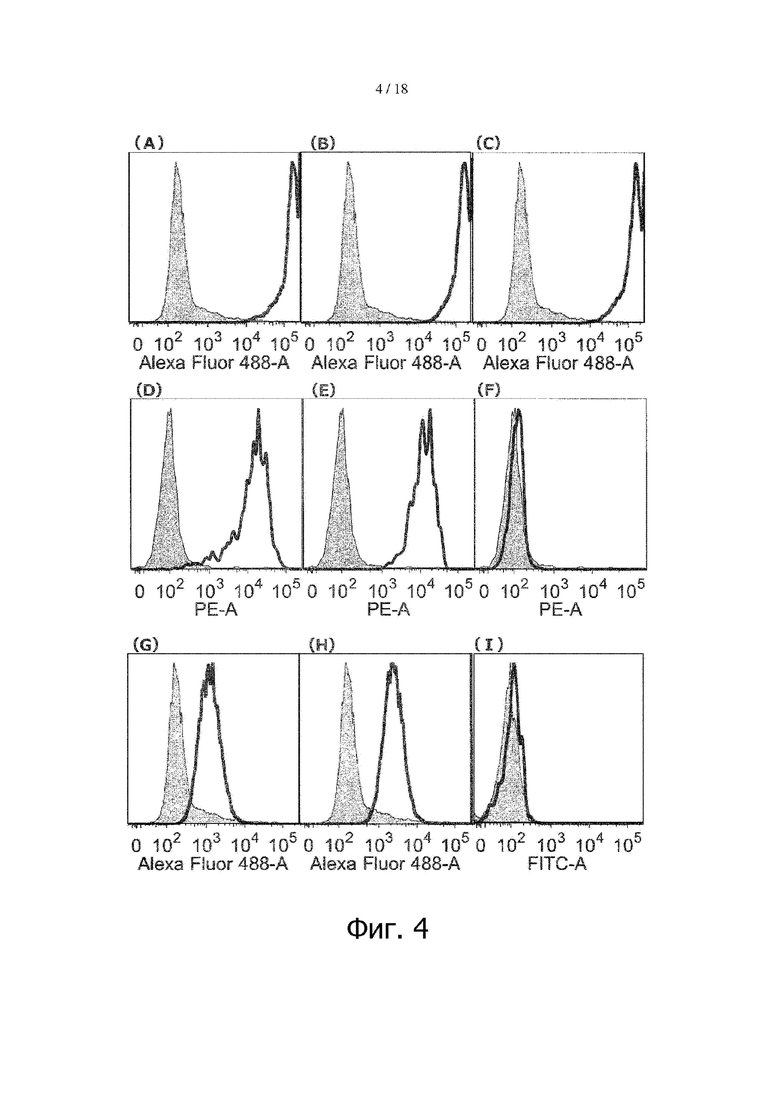
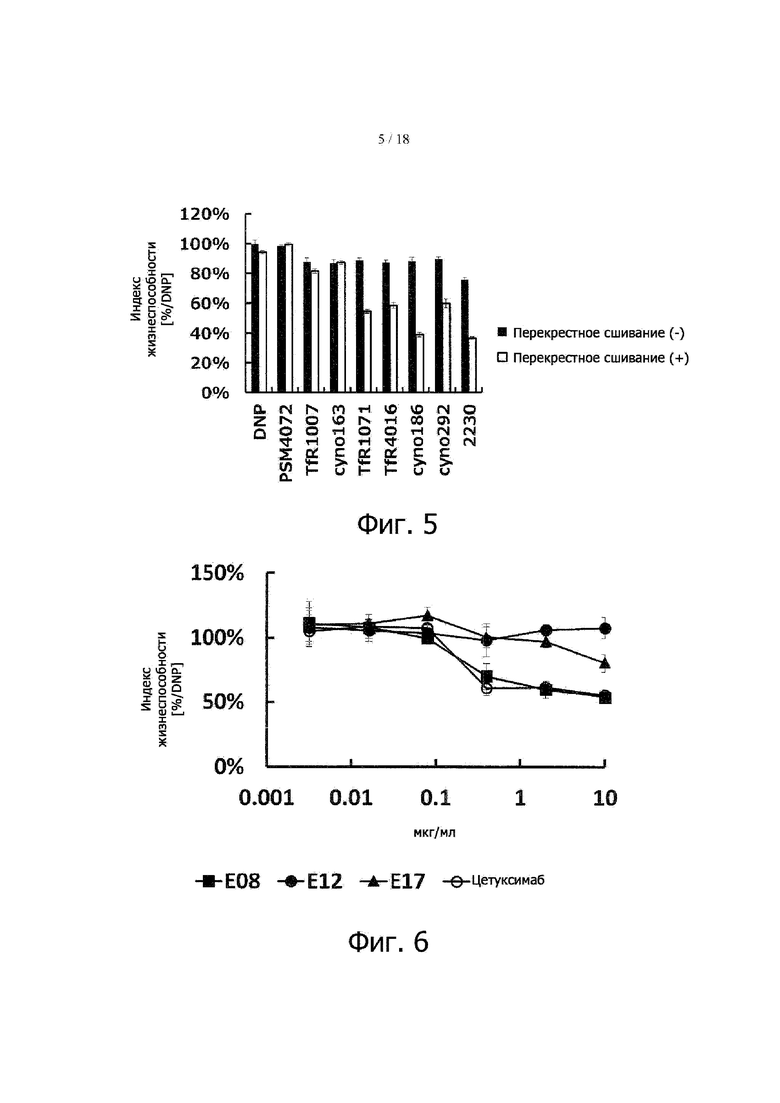

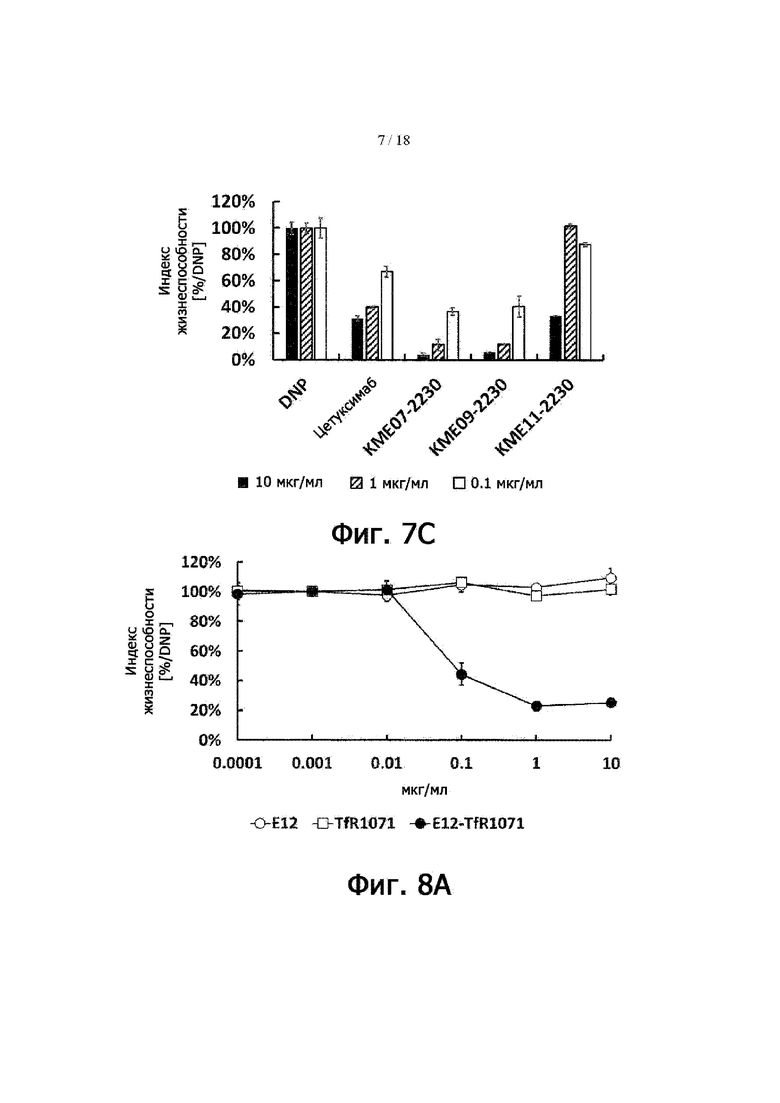
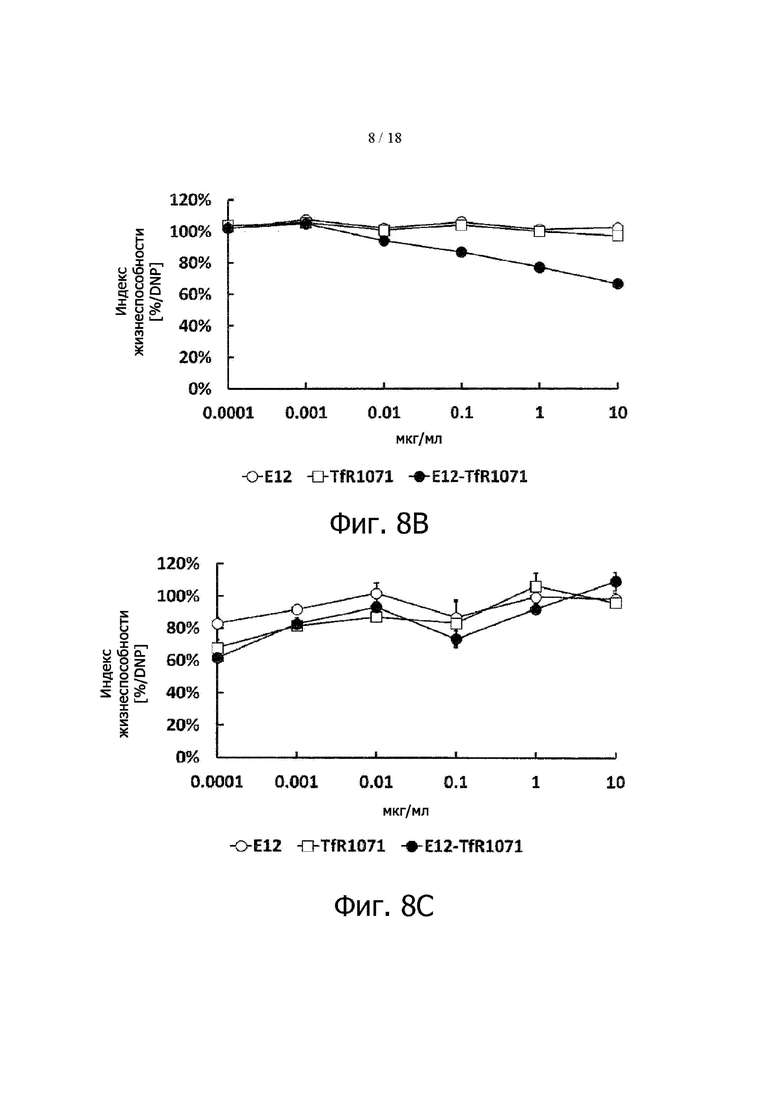
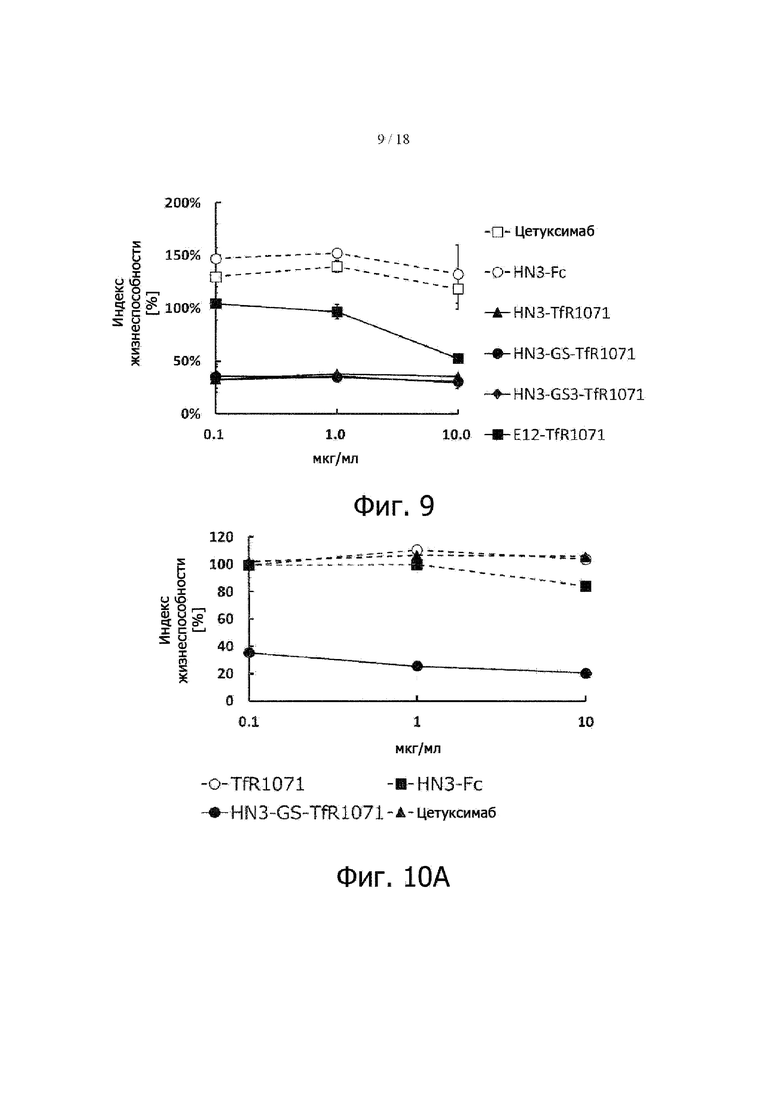
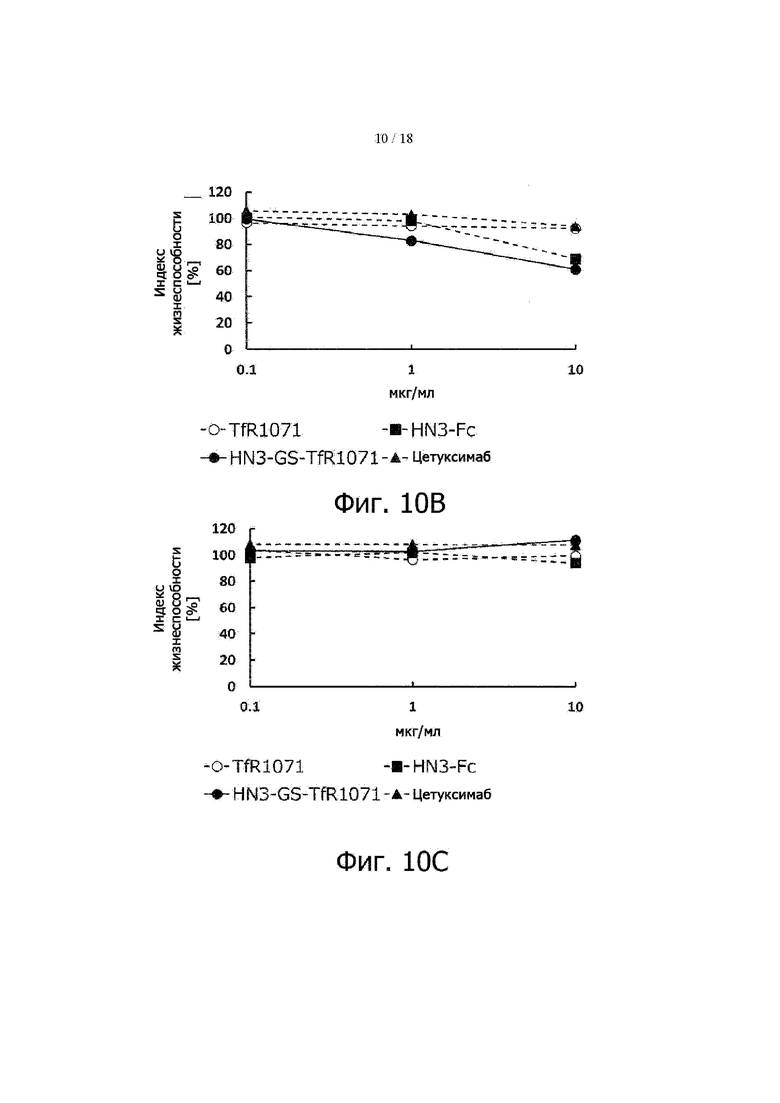
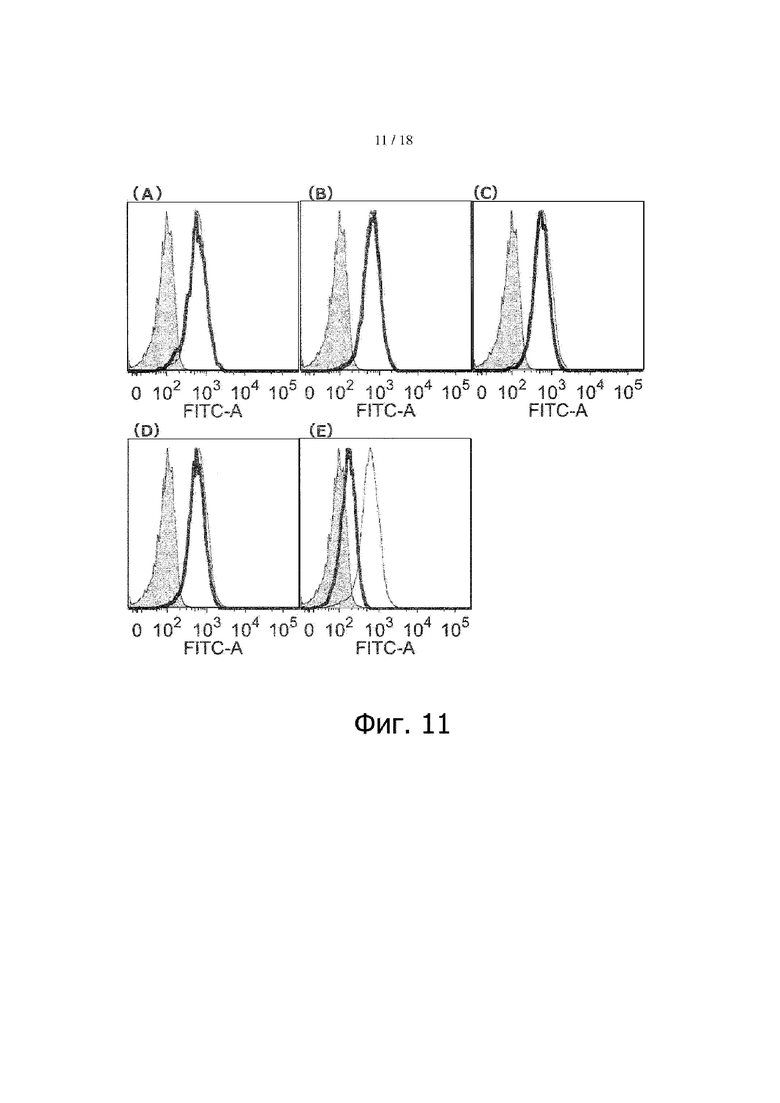
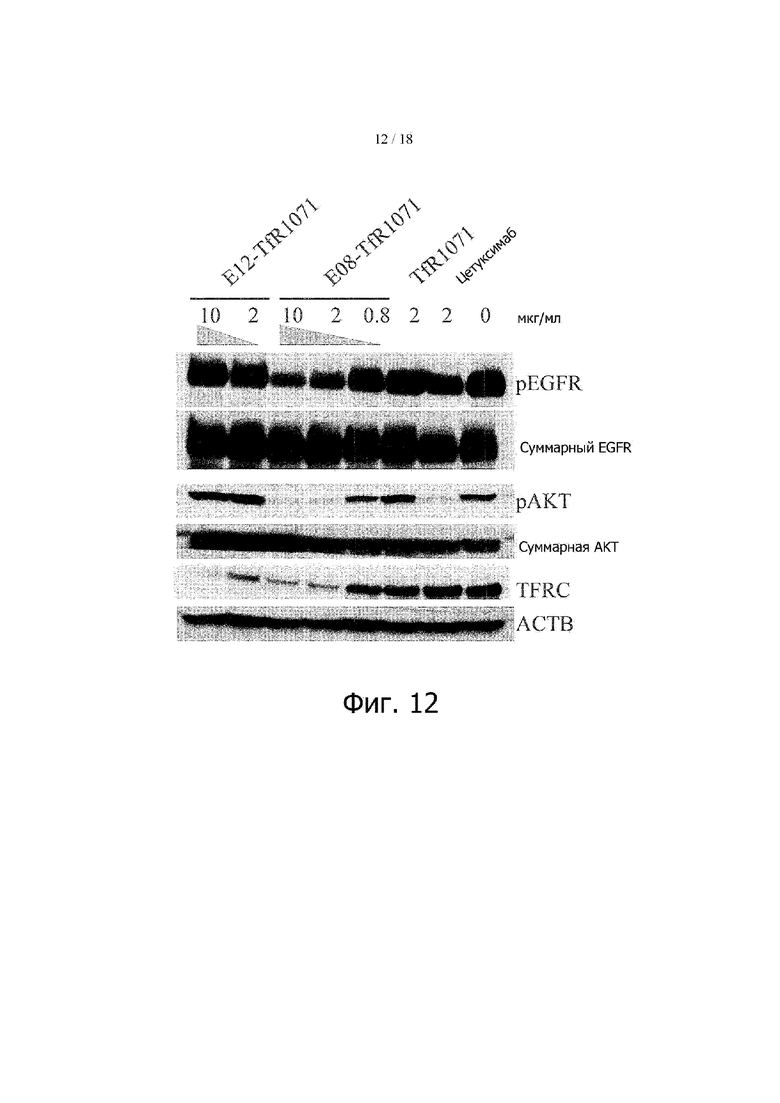
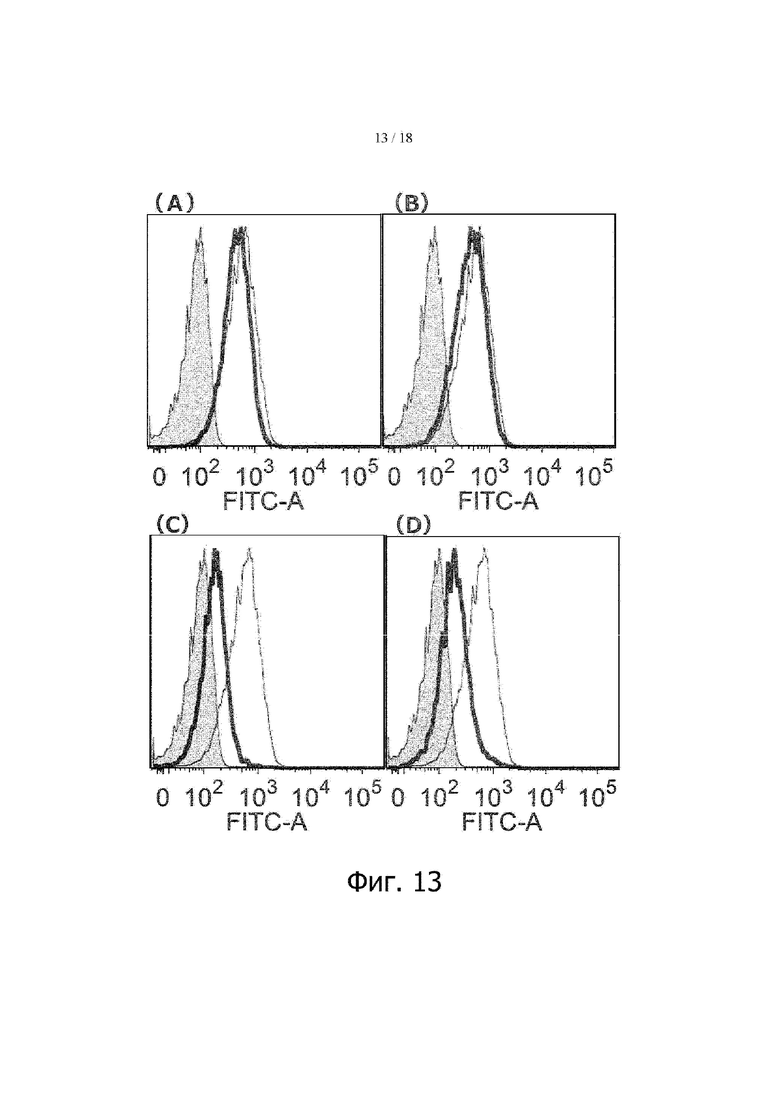

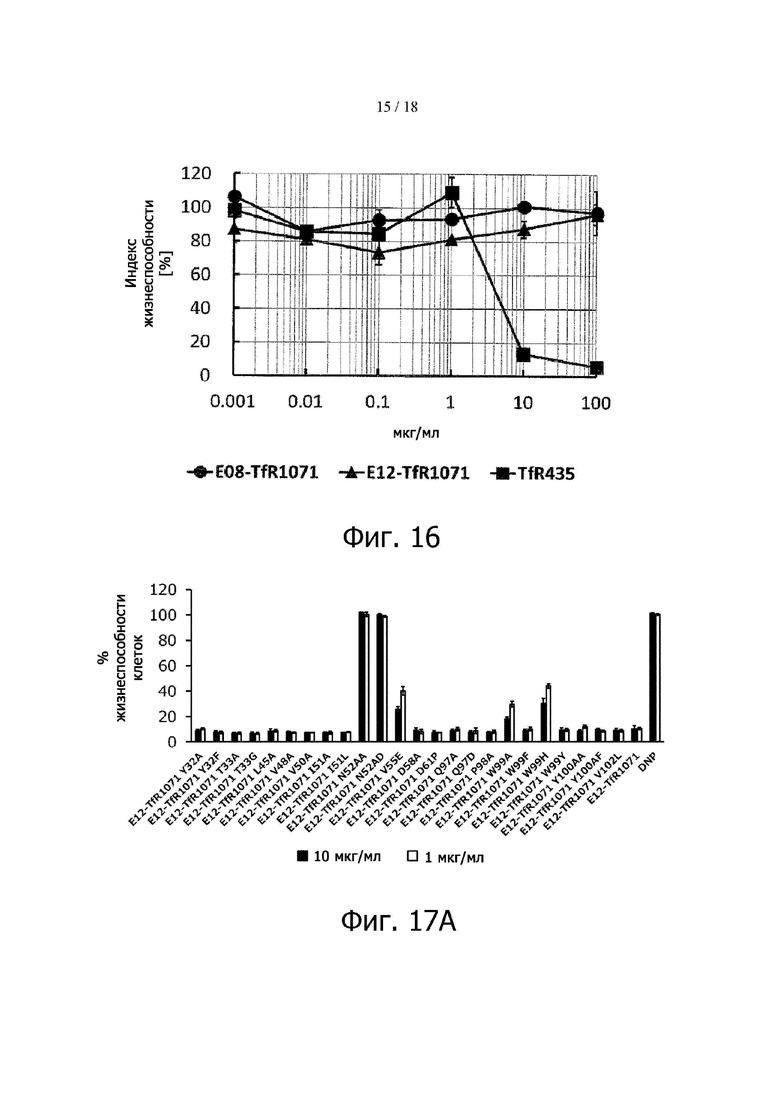
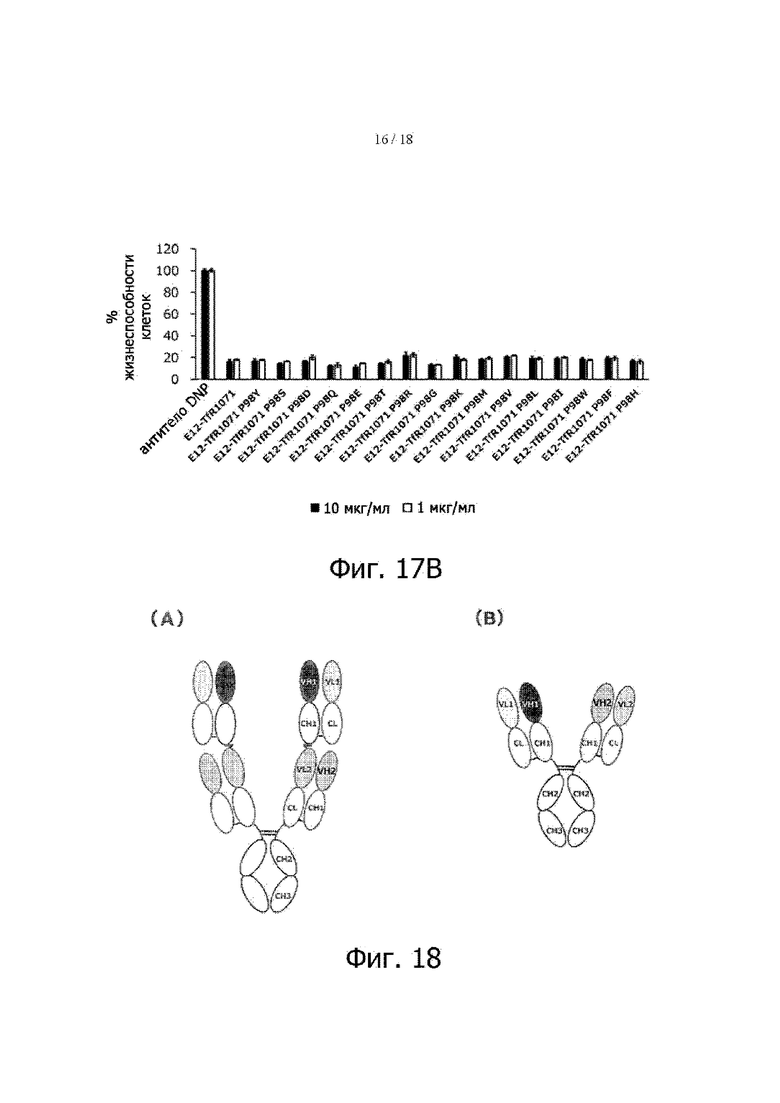
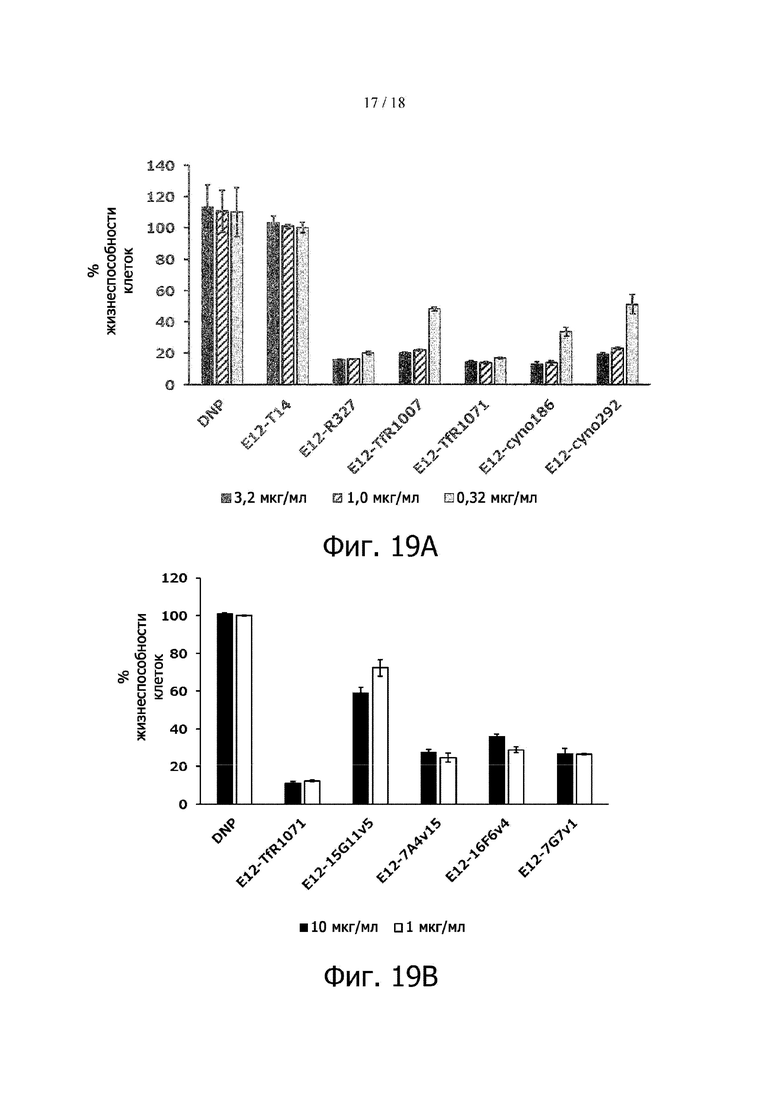
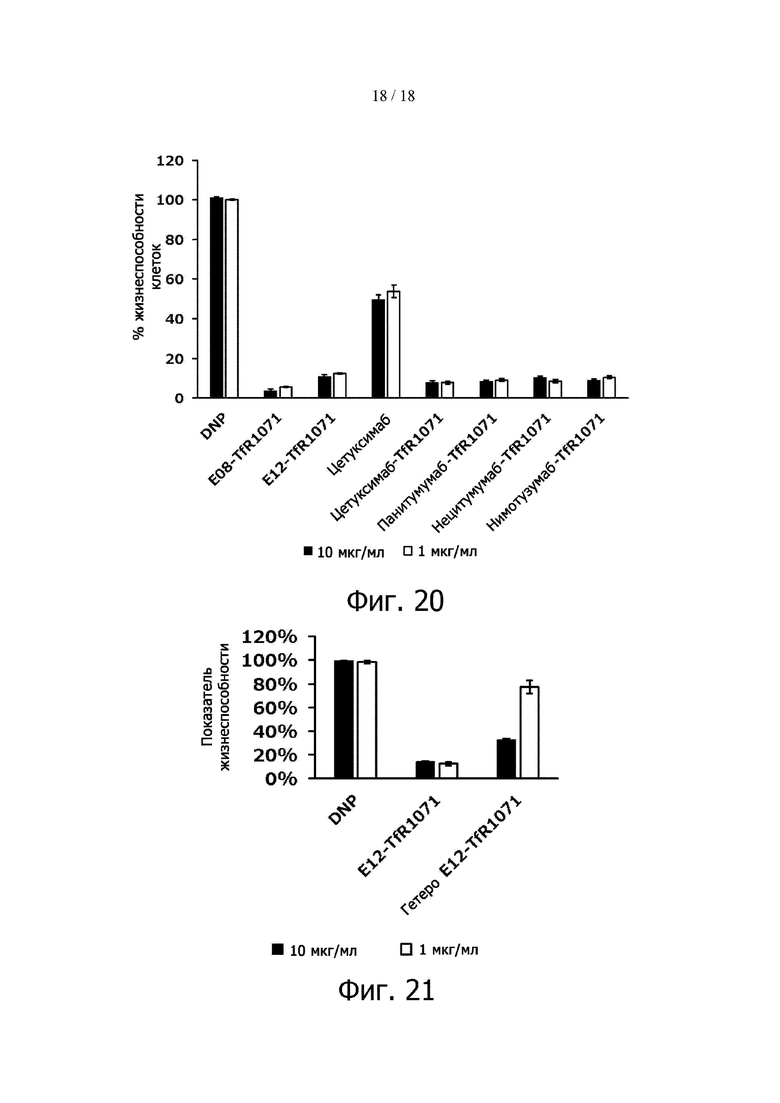
Изобретение относится к области биотехнологии, в частности к биспецифическому антителу или фрагменту этого биспецифического антитела, которые связываются с рецептором трансферрина (TfR) и с антигеном клеточной поверхности, а также к способу его получения. Также раскрыты ДНК, кодирующая вышеуказанное антитело или его фрагмент, а также вектор и трансформант, ее содержащие. Изобретение эффективно для лечения заболевания, связанного по меньшей мере с одним из человеческого TfR и EGFR, которое представляет собой раковое заболевание. 9 н. и 9 з.п. ф-лы, 21 ил., 7 табл., 20 пр.
1. Биспецифическое антитело или фрагмент этого биспецифического антитела, которые связываются с рецептором трансферрина (TfR) и с антигеном клеточной поверхности, включающие IgG часть, которая связывается с TfR, и полипептид N-концевой стороны, который связывается с антигеном клеточной поверхности, причем:
(i) указанный антиген клеточной поверхности представляет собой EGFR;
(ii) IgG часть включает одну вариабельную область тяжелой цепи (VH), выбранную из следующих (ai)-(ci), и вариабельную область легкой цепи (VL), имеющую CDR 1-3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 17-19 соответственно:
(ai) VH, имеющий CDR 1-3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 32-34 соответственно;
(bi) VH, имеющий CDR 1-3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 42-44 соответственно; и
(ci) VH, имеющий CDR 1-3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 47-49 соответственно;
(iii) полипептид N-концевой стороны представляет собой Fab-антитела против EGFR, где антитело включает VH, имеющий CDR 1-3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 60-62 соответственно, или SEQ ID NO: 65-67 соответственно, и VL, имеющий CDR 1-3, содержащие аминокислотные последовательности, представленные SEQ ID NO: 17-19 соответственно; и
(iv) указанный полипептид N-концевой стороны связан непосредственно или через линкерную структуру с N-концом тяжелой цепи указанной части IgG.
2. Биспецифическое антитело или фрагмент этого биспецифического антитела по п. 1, где указанное биспецифическое антитело бивалетно связывается как с TfR, так и с EGFR.
3. Биспецифическое антитело или фрагмент этого биспецифического антитела по п. 1 или 2, в котором часть IgG включает VH, содержащую аминокислотную последовательность, представленную любой из последовательностей SEQ ID NO: 31, 41 и 46, и VL, содержащую аминокислотную последовательность, представленную SEQ ID NO: 16.
4. Биспецифическое антитело или фрагмент этого биспецифического антитела по любому из пп. 1–3, в котором часть IgG содержит константную область тяжелой цепи аминокислотной последовательности, представленной SEQ ID NO: 84 или SEQ ID NO: 86.
5. Биспецифическое антитело или фрагмент этого биспецифического антитела по любому из пп. 1-4, в котором антитело против EGFR представляет собой антитело, включающее VH, содержащую аминокислотную последовательность, представленную SEQ ID NO: 59 или SEQ ID NO: 64, и VL, содержащую аминокислотную последовательность, представленную SEQ ID NO: 16.
6. Биспецифическое антитело или фрагмент этого биспецифического антитела по любому из пп. 1-5, в котором С-конец тяжелой цепи Fab непосредственно соединен с N-концом тяжелой цепи части IgG.
7. Биспецифическое антитело или фрагмент этого биспецифического антитела по любому из пп. 1-5, в котором С-конец тяжелой цепи Fab связан с N-концом тяжелой цепи части IgG посредством линкера.
8. Биспецифическое антитело или фрагмент этого биспецифического антитела по п. 7, в котором аминокислотная последовательность линкера состоит из всей аминокислотной последовательности шарнирного участка молекулы IgG или ее части.
9. Биспецифическое антитело или фрагмент этого биспецифического антитела по любому из пп. 1-8, в котором аминокислотная последовательность, из которой состоит легкая цепь в фрагменте Fab, и аминокислотная последовательность, из которой состоит легкая цепь в части IgG, одинаковые.
10. ДНК, кодирующая биспецифическое антитело или фрагмент этого биспецифического антитела по любому из пп. 1-9.
11. Экспрессионный рекомбинантный вектор, содержащий ДНК по п. 10.
12. Трансформант для получения биспецифического антитела или фрагмента этого биспецифического антитела по любому из пп. 1-9, содержащий рекомбинантный вектор по п. 11, где трансформант представляет собой клетку.
13. Способ получения биспецифического антитела или фрагмента этого биспецифического антитела по любому из пп. 1-9, включающий культивирование трансформанта по п. 12 в культуральной среде, обеспечивающей продуцирование трансформантом и накопление в культуре биспецифического антитела или фрагмента этого биспецифического антитела по любому из пп. 1-9, а также включающий выделение указанного биспецифического антитела или фрагмента этого биспецифического антитела из этой культуры.
14. Терапевтический агент, применяемый при заболевании, связанном по меньшей мере с одним из человеческого TfR или EGFR, причем указанный агент включает в качестве активного ингредиента биспецифическое антитело или фрагмент этого биспецифического антитела по любому из пп. 1-9, где заболевание, связанное по меньшей мере с одним из человеческого TfR или EGFR, представляет собой раковое заболевание.
15. Терапевтический способ лечения заболевания, связанного по меньшей мере с одним из человеческого TfR и EGFR, в котором используется биспецифическое антитело или фрагмент этого биспецифического антитела по любому из пп. 1-9, где заболевание, связанное по меньшей мере с одним из человеческого TfR или EGFR, представляет собой раковое заболевание.
16. Биспецифическое антитело или фрагмент этого биспецифического антитела по любому из пп. 1-9 для применения при лечении заболевания, связанного по меньшей мере с одним из человеческого TfR и EGFR, где заболевание, связанное по меньшей мере с одним из человеческого TfR или EGFR, представляет собой раковое заболевание.
17. Применение биспецифического антитела или фрагмента биспецифического антитела по любому из пп. 1-9 для получения терапевтического агента для лечения заболевания, связанного по меньшей мере с одним из человеческого TfR и EGFR, где заболевание, связанное по меньшей мере с одним из человеческого TfR или EGFR, представляет собой раковое заболевание.
18. Применение биспецифического антитела или фрагмента биспецифического антитела по любому из пп. 1-9 для получения терапевтического агента для лечения заболевания, связанного по меньшей мере с одним из TfR человека и EGFR, где заболевание, связанное по меньшей мере с одним из человеческого TfR или EGFR, представляет собой раковое заболевание.
| WO 2014189973 A2, 27.11.2014 | |||
| WO 2012075037 A1, 07.06.2012 | |||
| WO 2013177062 A2, 28.11.2013 | |||
| ULRICH BRINKMANN et al., The making of bispecific antibodies, 2017, mAbs, 9:2, 182-212 | |||
| RU 2016129624 A, 08.02.2018. |
Авторы
Даты
2023-12-28—Публикация
2019-12-27—Подача