ОБЛАСТЬ ТЕХНИКИ
Настоящее изобретение относится к нацеленным на опухоль биспецифическим агонистическим CD28-антигенсвязывающим молекулам, характеризуемым одновалентным связыванием с CD28, способам их получения, фармацевтическим композициям, содержащим эти молекулы, и их применению в лечении рака.
УРОВЕНЬ ТЕХНИКИ
Иммунотерапия рака становится все более эффективным вариантом терапии, которая может обеспечивать поразительные и длительные ответы при таких типах рака, как меланома, немелкоклеточный рак легкого и почечно-клеточная карцинома. Это главным образом обусловлено успехом нескольких блокаторов иммунных контрольных точек, включая анти-PD-l (например, Кейтруда, Merck; Opdivo, BMS), анти-CTLA-4 (например, Ервой, BMS) и анти-PD-L1 (например, Тецентрик, Roche). Эти агенты вероятно станут стандартом лечения для многих типов рака или основой комбинированной терапии, однако, такая терапия обеспечивает благоприятный эффект только для части пациентов (<25%). Кроме того, различные виды рака (рак предстательной железы, колоректальный рак, рак поджелудочной железы, саркомы, отличный от трижды негативного рак молочной железы и т.д.) проявляют первичную резистентность к этим иммуномодуляторам. В ряде сообщений указано, что отсутствие предсуществующих противоопухолевых Т-клеток способствует отсутствию или слабому ответу некоторых пациентов. В целом, несмотря на впечатляющие противораковые эффекты существующих вариантов иммунотерапии, существует очевидная медицинская потребность в решении проблем большой популяции раковых пациентов и в разработке вариантов терапии, целью которых является индукция и усиление новых опухолеспецифических Т-клеточных ответов.
CD28 является основным членом подсемейства ко стимулирующих молекул, характеризуемых наличием спаренных доменов V-группы суперсемейства иммуноглобулинов (IgSF), присоединенных к одинарным трансмембранным доменам и цитоплазматическим доменам, которые содержат критически важные сигнальные мотивы (Carreno and Collins, 2002). Другие члены этого подсемейства включают ICOS, CTLA-4, PD1, PD1H, TIGIT и BTLA (Chen and Flies, 2013). Экспрессия CD28 ограничена Т-клетками и преобладает на всех наивных и большинстве контактировавших с антигеном подгрупп, включая те, которые экспрессируют PD-1 или CTLA-4. CD28 и CTLA-4 являются высокогомологичными и конкурируют за связывание с одними и теми же В7-молекулами CD80 и CD86, которые экспрессируются на дендритных клетках, В-клетках, макрофагах и опухолевых клетках (Linsley et al., 1990). Более высокая аффинность CTLA-4 в отношении семейства лигандов В7 позволяет CTLA-4 превосходить CD28 в отношении связывания лиганда и подавлять ответы эффекторных Т-клеток (Engelhardt et al., 2006). В противоположность этому, было показано, что PD-1 ингибирует сигнализацию CD28 путем частичного дефосфорилирования цитоплазматического домена CD28 (Hui et al., 2017). Осуществляемое CD80 или CD86 лигирование CD28 на поверхности профессиональных антигенпрезентирующих клеток обязательно необходимо для функционального примирования наивных Т-клеток de novo, последующих клональной экспансии, выработки цитокинов, лизиса клеток-мишеней и формирования долгосрочной памяти. Связывание лигандов CD28 также способствует экспрессии индуцибельных костимулирующих рецепторов, таких как ОХ-40, ICOS и 4-1 ВВ (обзор в Acuto and Michel, 2003). Было показано, что после лигирования CD28 дисульфид-связанный гомодимер, проксимальный к мембране мотив YMNM и дистальный мотив PYAP образуют комплекс с несколькими киназами и адапторными белками (Boomer and Green, 2010). Эти мотивы важны для индукции транскрипции IL2, которая опосредована CD28-зависимой активацией транскрипционных факторов семейств NFAT, АР-1 и NFκB (Fraser et al., 1991) (June et al., 1987) (Thompson et al., 1989). Однако в цитоплазматическом домене CD28 были обнаружены дополнительные сайты фосфорилирования и убиквитинирования с плохо изученными характеристиками. Согласно обзору (Esensten et al., 2016) инициируемые CD28 пути играют критическую роль в стимуляции пролиферации и эффекторной функции традиционных Т-клеток. Лигирование CD28 также стимулирует противовоспалительную функцию регуляторных Т-клеток. CD28 костимулирует Т-клетки частично путем усиления сигналов от Т-клеточного рецептора, но также было показано, что он опосредует уникальные сигнальные события (Acuto and Michel, 2003; Boomer and Green, 2010; June et al., 1987). Сигналы, специфически инициируемые CD28, регулируют многие важные аспекты функции Т-клеток, включая фосфорилирование и другие посттрансляционные модификации последующих белков (например, P13K-опосредованное фосфорилирование), транскрипционные изменения (например, экспрессию Bcl-xL), эпигенетические изменения (например, промотор IL-2), ремоделирование цитоскелета (например, ориентации центра организации микротрубочек) и изменения гликолитической скорости (например, гликолитического потока). Мыши с дефицитом CD28 имели сниженные ответы на инфекционные патогены, аллотрансплантатные антигены, болезнь «трансплантат против хозяина», контактную гиперчувствительность и астму (Acuto and Michel, 2003). Отсутствие CD28-опосредованной костимуляции приводит к снижению пролиферации Т-клеток in vitro и in vivo, к сильному ингибированию образования зародышевого центра и переключения изотипов и классов иммуноглобулина, снижению дифференцировки Т-хелперных (Th) клеток и экспрессии цитокинов Th2 типа. Также наблюдается отрицательное влияние на CD4-зависимые ответы цитотоксических CD8+ Т-клеток. Важно то, что CD28-дефицитные наивные Т-клетки демонстрировали сниженный пролиферативный ответ, в частности на более низкие концентрации антигена. Накапливающиеся литературные данные свидетельствуют в пользу идеи, что задействование CD28 на Т-клетках имеет противоопухолевый потенциал. Последние данные демонстрируют, что противораковые эффекты ингибиторов контрольных точек PD-L1/PD-1 и CTLA-4 зависят от CD28 (Kamphorst et al., 2017; Tai et al., 2007). Клинические исследования, в которых изучали терапевтические эффекты блокирования CTLA-4 и PD-1, показали исключительно многообещающие результаты у пациентов с распространенной меланомой и другими видами рака. Помимо этого, инфузия генетически сконструированных Т-клеток, экспрессирующих искусственные химерные Т-клеточные рецепторы, содержащие внеклеточный антиген-распознающий домен, слитый с внутриклеточными сигнальными доменами ТКР (CD3z) и внутриклеточными костимулирующими доменами (доменами CD28 и/или 4-1ВВ), продемонстрировала высокие уровни и продолжительность ответа при В-клеточных раках и других видах раков.
Агонистические антитела к CD28 можно разделить на две категории: (i) суперагонистические антитела к CD28 и (ii) стандартные агонистические антитела к CD28. Обычно для активации Т-клеток необходимо как задействование Т-клеточного антигенного рецептора (ТКР, сигнал 1), так и костимулирующая сигнализация CD28 (сигнал 2). Суперагонисты CD28 (CD28SA) представляют собой CD28-специфические моноклональные антитела, которые способны автономно активировать Т-клетки без явного задействования Т-клеточного рецептора (Hűnig, 2012). У грызунов CD28SA активирует стандартные и регуляторные Т-клетки. Антитела к CD28SA терапевтически эффективны во многих моделях аутоиммунности, воспаления и трансплантации. Однако результатом исследования фазы I человеческого антитела к CD28SA TGN1412 в 2006 г. стал опасный для жизни цитокиновый шторм. Последующие исследования подтвердили, что токсичность была вызвана ошибками в дозировке вследствие различий в восприимчивости к CD28 человеческих Т-клеток и Т-клеток доклинических животных моделей. Сейчас TGN1412 проходит переоценку в открытом многоцентровом исследовании с повышением дозы среди пациентов с РА и пациентов с метастатическими или неоперабельными распространенными солидными злокачественными образованиями. Стандартные агонистические антитела к CD28, такие как клон 9.3, имитируют природные лиганды CD28 и являются единственными способными усиливать активацию Т-клеток в присутствии сигнала Т-клеточного рецептора (сигнал 1). Опубликованные результаты показывают, что связывающий эпитоп антитела имеет основное влияние на то, является ли агонистическое антитело суперагонистом или стандартным агонистом (Beyersdorf et al., 2005). Суперагонистическое TGN1412 связывается с латеральным мотивом CD28, тогда как стандартная агонистическая молекула 9.3 связывается вблизи связывающего лиганд эпитопа. Как следствие наличия разных связывающих эпитопов суперагонистическое и стандартное агонистическое антитела отличаются по своей способности образовывать линейные комплексы молекул CD28 на поверхности Т-клеток. А именно, TGN1412 способно эффективно образовывать линейные комплексы с CD28, что предположительно приводит к агрегации сигнальных компонентов, которых достаточно для преодоления порога активации Т-клеток. С другой стороны, стандартный агонист 9.3 приводит к образованию комплексов, которые не являются линейными по структуре. Ранее были опубликованы данные (Otz et al., 2009) о попытке преобразования стандартных агонистических связывающих молекул на основании клона 9.3 с использованием рекомбинантного биспецифического одноцепочечного антитела, направленного на ассоциированный с меланомой протеогликан и CD28. Сообщалось, что описанное биспецифическое одноцепочечное антитело проявляло «сверхагонистическую» активность несмотря на использование стандартной агонистической CD28-связывающей молекулы 9.3 благодаря свойственной биспецифическим одноцепочечным антителам тенденции образовывать мультимерные конструкции.
Было обнаружено, что лучшая активация Т-клеток достигается, когда ограниченные количества анти-CD3 биспецифических антител, т.е. Т-клеточных биспецифических антител (ТСВ, англ. «Т cell bispecific antibody»), таких как СЕА-ТСВ, комбинируют с агонистическими анти-CD28 молекулами. Учитывая, что CD28 экспрессируется на исходном уровне на Т-клетках при различных опухолевых показаниях (Lavin et al., 2017; Tirosh et al., 2016, Zheng et al., 2017), а активация сигнализации CD28 усиливает сигналы Т-клеточных рецепторов, ожидается, что комбинация молекулы ТСВ с нацеленной на опухоль молекулой CD28 будет действовать синергетически и индуцировать сильные и продолжительные противоопухолевые ответы. Таким образом, в данном документе мы описываем новые нацеленные на опухоль агонистические к CD28 молекулы, которые демонстрируют синергизм с ТСВ и для которых необходима одновалентность связывания CD28 для строгой зависимости от опухолевой мишени в присутствии сигналов ТСВ.
Иммунотерапия солидных опухолей
Лечение солидных опухолей является текущей задачей, прогресс решения которой за последние годы был небольшим. Как правило, лечение является комбинацией хирургии и химиотерапии и/или лучевой терапии. Хотя за последнее время было разработано немало новых режимов лечения, все еще существует потребность в дополнительных усовершенствованиях для повышения уровней выживаемости пациентов, страдающих солидными опухолями, и улучшения качества их жизни. Солидные опухоли редко экспрессируют один опухолеспецифический антиген. Для большинства солидных опухолей более распространенным является наличие опухолеассоциированного антигена (ОАА), который в повышенном количестве представлен в опухолях, но также на очень низких уровнях экспрессируется в нормальных тканях. ОАА предпочтительно экспрессируется на поверхности клеток солидной опухоли или на клетках опухолевой стромы. Это справедливо для многих часто служащих мишенями ОАА в случае солидных опухолей, включая белок активации фибробластов (FAP), карциноэмбриональный антиген (СБА), рецептор фолиевой кислоты альфа (FolR1), ассоциированный с меланомой хондроитинсульфата протеогликан (MCSP), рецептор эпидермального фактора роста (EGFR), рецептор 2 эпидермального фактора роста человека (HER2) и p95HER2. Дополнительные ОАА включают HER3, ЕрСАМ, TPBG (5Т4), мезотелин, MUC1 и PSMA. Таким образом, биспецифическая агонистическая CD28-антигенсвязывающая молекула, содержащая антигенсвязывающий домен, который специфически связывается с опухолеассоциированным антигеном, будет нацелена главным образом на поверхность опухоли или микроокружение опухоли и будет специфически активировать Т-клетки вблизи опухоли, при этом системной активации можно избежать.
Обоснование нацеливания агонизма CD28 на В-клеточные злокачественные образования
Неходжкинская лимфома (НХЛ) является одной из основных причин смерти от рака в Соединенных Штатах и в Европе. Фолликулярная лимфома (ФЛ) характеризуется медленным прогрессированием и имеет низкую скорость развития с медианной выживаемостью от 8 до 10 лет; пациенты на стадиях распространенного клинического заболевания обычно неизлечимы. Аналогично, у 2% - 3% пациентов в год фенотип ФЛ может трансформироваться в агрессивную крупноклеточную лимфому, что является критическим событием в ходе заболевания и связано с повышенной смертностью от лимфомы. Мантийноклеточная лимфома и диффузная В-крупноклеточная лимфома (ДВКЛ) являются более агрессивными, и при отсутствии лечения медианный уровень выживаемости составляет всего 6 месяцев. Отсутствие терапевтических результатов для многих пациентов как с вялотекущим, так и с агрессивным подтипами НХЛ, остается неудовлетворенной медицинской потребностью несмотря на значительные успехи иммунотерапевтических средств, которые позволили продлить время выживаемости без прогрессирования. На протяжении последних нескольких лет наблюдали значительно продленную выживаемость при ДВКЛ, в частности при добавлении анти-CD20 моноклонального антитела ритуксимаба (Ритуксан®, MabThera®) в интенсивные цитотоксические химиотерапевтические схемы. Однако несмотря на то, что стандартное лечение ранее нелеченной ДВКЛ направлено на полное излечение, у большинства пациентов в конечном итоге случается рецидив. Аналогично, распространенная ФЛ остается в целом неизлечимой современными стандартами лечения и характеризуется повторными рецидивами и постепенно укорачивающимися периодами ремиссии. На сегодняшний день многие моноклональные антитела нового поколения находятся на разных доклинических и клинических фазах оценки для дополнительного улучшения результатов пациентов с НХЛ и преодоления механизмов резистентности к ритуксимабу. Химиотерапия высокими дозами с поддержкой аутологичных стволовых клеток или трансплантацией аллогенных стволовых клеток обеспечивает полное излечение только для малой части (10%) пациентов с рецидивирующей/рефрактерной (р/р) ДВКЛ и ассоциируется со значительным уровнем связанной с лечением смертности. Другие подходы для лечения НХЛ, находящиеся на данный момент на стадии разработки, включают молекулярные нацеленные соединения, такие как венетоклакс и ВЕТ-ингибиторы. Недавно одобренные новые агенты включают леналидомид, иделалисиб и копанлисиб. Т-клеточная терапия с химерным антигенным рецептором (CAR) была одобрена для лечения агрессивных форм р/р В-НХЛ, но эта терапия доступна только в ограниченных условиях и может быть связана с фатальными неврологическими событиями и синдромом высвобождения цитокинов (СВЦ). Биспецифические конструкции антител, перенаправляющие лизис цитотоксическими клетками на злокачественные В-клетки, на сегодняшний день находятся на стадии разработки и успели продемонстрировать очень многообещающую эффективность против НХЛ. В будущем для НХЛ предусмотрены варианты лечения без химиотерапии и, вероятно, они будут основаны на биспецифических антителах или Т-клетках с химерным антигенным рецептором (CAR Т-клетки). Агонист CD28, направленный против поверхностного антигена В-клеток, в комбинации с иммунотерапией должен повышать уровни выживаемости и/или излечения для пациентов с В-клеточными злокачественными образованиями, не ухудшая качество их жизни.
Поверхностные антигены В-клеток в качестве мишени при В-клеточных злокачественных образованиях
ОАА, связанными с В-клеточными злокачественными образованиями, являются поверхностные антигены В-клеток. Человеческий антиген CD19 представляет собой 95 кДа трансмембранный гликопротеин, принадлежащий к суперсемейству иммуноглобулинов. CD19 классифицируется как трансмембранный белок I типа с одним трансмембранным доменом, цитоплазматическим С-концом и внеклеточным Ν-концом. В нормальных клетках он является наиболее повсеместно экспрессируемым белком в линии дифференцировки В-лимфоцитов. Экспрессия CD19 сохраняется в клетках В-линии дифференцировки, которые подверглись неопластической трансформации, и следовательно, CD19 можно применять при диагностике лейкозов и лимфом, используя моноклональные антитела (mAb) и проточную цитометрию, и то же касается антигена CD20. Поскольку лейкозы и лимфомы В-линии дифференцировки редко утрачивают экспрессию CD19 и поскольку он не экспрессируется в плюрипотентных стволовых клетках, он стал мишенью для ряда иммунотерапевтических агентов, включая иммунотоксины. CD79 является сигнальным компонентом В-клеточного рецептора и состоит из ковалентного гетеродимера, содержащего CD79a (Igα, mb-1) и CD79b (Igβ, B29). Каждый из CD79a и CD79b содержит внеклеточный домен иммуноглобулина (Ig), трансмембранный домен и внутриклеточный сигнальный домен, домен иммунорецепторного тирозинового активирующего мотива (ITAM), как и другие сигнальные белки, такие как CD3 или активирующий Рсу-рецептор. Следовательно, CD79a и CD79b являются трансмембранными белками, которые составляют сигнальные субъединицы В-клеточного рецептора (BCR). CD79b представляет собой 39 кДа белок, экспрессируемый исключительно на В-клетках, и вместе с CD79a инициирует каскад сигнальной трансдукции после BCR, что приводит к интернализации комплекса BCR, его транслокации в эндосомы и презентации антигена. В В-клетках антиген-индуцированная кластеризация BCR инициирует фосфорилирование тирозина в ITAM CD79a и CD79b Src-каназами. Это приводит к рекрутированию и активации комплексов эффекторных молекул, принадлежащих к сигнальному каскаду BCR, включая наиболее заметные SYK и BLNK. Далее рекрутирование PLCg2, Btk и ERK облегчает приток кальция и активирует В-клетки, которые после этого готовы принимать дополнительные коактивирующие сигналы, которые будут обуславливать их пролиферацию и дифференцировку в клетки памяти или эффекторные клетки. Во время этого процесса В-клетки становятся активными АПК и высвобождают цитокины, которые могут влиять на результат и качество иммунного ответа. Помимо своей роли в сигнализации BCR, субъединицы CD79 также важны для транспорта и отображения мембраносвязанного Ig из эндоплазматического ретикулума на поверхности клетки. Средняя поверхностная экспрессия CD79b в НХЛ сходна с таковой на нормальных В-клетках, но с более широким диапазоном. С учетом экспрессии CD79b, целесообразно получать терапевтические антитела к антигену CD79b, которые создают минимальную или отсутствующую антигенность при введении пациентам, в особенности в случае хронического лечения.
Было обнаружено, что лучшая активация Т-клеток достигается, когда ограниченные количества анти-CD3 биспецифических антител, т.е. Т-клеточных биспецифических антител (ТСВ), таких как, например биспецифическое антитело CD20/CD3, комбинируют с агонистическими анти-CD28 молекулами. Учитывая, что CD28 экспрессируется на исходном уровне на Т-клетках при различных опухолевых показаниях (Lavin et al., 2017; Tirosh et al., 2016, Zheng et al., 2017), а активация сигнализации CD28 усиливает сигналы Т-клеточных рецепторов, ожидается, что комбинация Т-клеточных биспецифических антител с биспецифическими агонистическими CD28-антигенсвязывающими молекулами, нацеленными на поверхностный антиген В-клеток, будет действовать синергетически и индуцировать сильные и продолжительные противоопухолевые ответы. Таким образом, в данном документе мы описываем новые биспецифические агонистические CD28-антигенсвязывающие молекулы, нацеленные на поверхностный антиген В-клеток, которые демонстрируют синергизм с ТСВ и для которых необходима одновалентность связывания CD28 для строгой зависимости от опухолевой мишени в присутствии сигналов ТСВ.
Иммунотерапия при множественной миеломе
Поражая ~ 75000 новых пациентов каждый год в ЕС и США, множественная миелома (ММ) является одним из наиболее распространенных гематологических злокачественных образований с остающейся острой неудовлетворенной медицинской потребностью. Множественная миелома характеризуется терминально дифференцированными плазматическими клетками, которые секретируют нефункциональные моноклональные иммуноглобулины. В краткосрочной перспективе иммуномодулирующие лекарственные средства, такие как леналидомид и помалидомид, и ингибиторы протеасом, такие как карфилзомиб и бортезомиб, могут оставаться основой терапии 1-ой линии для множественной миеломы (Moreau et al, 2016). Однако эти лекарственные средства специфически не нацелены на патологические опухолевые клетки, например, патологические плазматические клетки (ПК). Были предприняты попытки в направлении избирательного истощения плазматических клеток при множественной миеломе. Отсутствие поверхностных белков, которые бы служили специфическими маркерами плазматических клеток, тормозит разработку антител или клеточной терапии для множественной миеломы. На сегодня существует всего несколько случае успешных биологических препаратов, включая даратумумаб (анти-CD38) и элотузумаб (анти-CD319), с оговоркой, что оба антигена также экспрессируются в других нормальных тканях, включая гемопоэтические линии дифференцировки и иммунные эффекторные клетки, что может ограничивать их долгосрочное клиническое применение. Антиген созревания В-клеток (ВСМА), трансмембранный гликопротеин в суперсемействе рецепторов фактора некроза опухолей 17 (TNFRSF17), экспессируется на значительно больших уровнях во всех клетках ММ пациента, но не в других нормальных тканях за исключением нормальных плазматических клеток. Т-клетки с ВСМА-химерным антигенным рецептором (CAR) уже продемонстрировали значительную клиническую активность у пациентов с РРММ, которые прошли по меньшей мере три предыдущих курса лечения, включающих ингибитор протеасом и иммуномодулирующий агент. Дополнительные схемы, включая конъюгат анти-ВСМА лекарственное средство, также позволили достичь значительных клинических ответов у пациентов, которые оказались невосприимчивыми к по меньшей мере трем предыдущим линиям терапии, включающим анти-CD38 антитело, ингибитор протеасом и иммуномодулирующий агент (Cho et al, 2018). Одна из проблем, например нацеленной на ВСМА или CD38 терапии, состоит в наличии высоких уровней растворимых ВСМА или CD38 в сыворотке пациентов с ММ, что может снижать количество активного лекарственного средства в организме пациента. Альтернативой могут быть новые мишени, такие как сопряженный с G-белком рецептор, класс С, группа 5, член D (GPRC5D), который дифференциально экспрессируется плазматическими клетками при множественной миеломе и плазматическими клетками от здоровых доноров и не имеет растворимой формы. Сообщалось, что GPRC5D связан с прогнозом и опухолевой нагрузкой у пациентов с множественной миеломой (Atamaniuk, J. et al., 2012; и Cohen, Y., et al., 2013). GPRC5D представляет собой орфанный рецептор с отсутствием известных лигандов и по большей части неизвестной биологией у человека в целом и при раке в частности. Кодирующий GPRC5D ген, картированный на хромосоме 12р13.3, содержит три экзона и занимает около 9,6 т.о. (Brauner-Osborne, Η. et al. 2001). Большой первый экзон кодирует семиспиральный трансмембранный домен. Было показано, что GPRC5D участвует в образовании кератина в волосяных фолликулах у животных (Gao, Υ. et al., 2016, и Inoue, S. et al., 2004). В WO 2018/017786 A2 описаны GPRC5D-специфические антитела или антигенсвязывающие фрагменты.
Обоснование для нацеливания агонизма CD28 на патологические плазматические клетки при множественной миеломе
Агонизм CD28 при множественной миеломе может оказывать разные биологические функции на иммунные соответствующие плазматические клетки ММ. Хотя ожидается, что активация Т-клеток посредством CD28 будет обуславливать противоопухолевые ответы, агонизм CD28 на клетках ММ опосредует способствующую выживаемости сигнализацию посредством регуляции PI3K/Akt, FoxO3a и Bimm, которые в свою очередь были описаны как индуцирующие химиотерапевтическую резистентность при множественной миеломе (Murray Μ.Ε. et al, 2014). Было описано, что сверхэкспрессия CD28 на плазматических клетках впервые диагностированной множественной миеломы коррелирует с плохим клиническим результатом (Bahlis et al., 2007). Однако, хотя активация CD28 повышает выживаемость клеток миеломы, его активация ингибирует пролиферацию клеток миеломы. Агонистическое воздействие на CD28 в присутствии сильного опосредованного иммунными клетками ответа, такого как биспецифическая активация Т-клеток, может дополнительно стимулировать эффективные противоопухолевые ответы. В данном документе мы предлагаем биспецифические агонистические CD28-антигенсвязывающие молекулы, которые специфически связывают поверхностный антиген клеток множественной миеломы (ММ) человека. В частности, биспецифические агонистические CD28-антигенсвязывающие молекулы в соответствии с изобретением, нацеленные на ОАА, выбранные из ВСМА, CD38 и GPRC5D, и CD28, экспрессируемый на Т-клетках, потенциально способны лечить множественную миелому как отдельный агент или в комбинации с другими агентами, такими как Т-клеточные биспецифические антитела (ТСВ), нацеленные на поверхностный антиген клеток ММ человека.
КРАТКОЕ ОПИСАНИЕ СУЩНОСТИ ИЗОБРЕТЕНИЯ
В настоящем изобретении описаны нацеленные на опухоль биспецифические агонистические CD28-антигенсвязывающие молекулы, которые обеспечивают опухоль-зависимую активацию Т-клеток и уничтожение опухолевых клеток без необходимости образования мультимеров. Биспецифические CD28-антигенсвязывающие молекулы по настоящему изобретению характеризуются одновалентным связыванием с CD28 и тем, что они содержат по меньшей мере один антигенсвязывающий домен, способный к специфическому связыванию с опухолеассоциированным антигеном (таким как белок активации фибробластов (FAP) или карциноэмбриональный антиген (СЕА), CD19 или GPRC5D). Кроме того, они содержат Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию. Следовательно, устраняется опосредованное Fc-рецептором перекрестное связывание, а опухолеспецифическая активация обеспечивается перекрестным связыванием посредством связывания по меньшей мере одного антигенсвязывающего домена, способного к специфическому связыванию с опухолеассоциированным антигеном, со своим антигеном.
Таким образом, в изобретении предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, характеризующаяся одновалентным связыванием с CD28, содержащая
(а) один антигенсвязывающий домен, способный к специфическому связыванию с CD28,
(б) по меньшей мере один антигенсвязывающий домен, способный к специфическому связыванию с опухолеассоциированным антигеном, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула по определению ниже, в которой Fc-домен принадлежит IgG, в частности представляет собой Fc-домен IgG1 или Fc-домен IgG4. В одном конкретном аспекте Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, представляет собой Fc-домен IgG1. В одном аспекте Fc-домен содержит аминокислотные замены L234A и L235A (нумерация в соответствии с индексом EU по Kabat). В одном аспекте Fc-домен принадлежит подклассу IgG1 человека и содержит аминокислотные мутации L234A, L235A и P329G (нумерация в соответствии с индексом EU по Kabat).
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула по определению выше в данном документе, в которой по меньшей мере один антигенсвязывающий домен, способный к специфическому связыванию с CD28, содержит
(i) вариабельную область тяжелой цепи (VHCD28), содержащую определяющую комплементарность область тяжелой цепи CDR-H1 с SEQ ID NO: 36, CDR-H2 с SEQ ID NO: 37 и CDR-H3 с SEQ ID NO: 38, и вариабельную область легкой цепи (VLCD28), содержащую определяющую комплементарность область легкой цепи CDR-L1 с SEQ ID NO: 39, CDR-L2 с SEQ ID NO: 40 и CDR-L3 с SEQ ID NO: 41; или
(ii) вариабельную область тяжелой цепи (VHCD28), содержащую CDR-H1 с SEQ ID NO: 20, CDR-H2 с SEQ ID NO: 21 и CDR-H3 с SEQ ID NO: 22, и вариабельную область легкой цепи (VLCD28), содержащую CDR-L1 с SEQ ID NO: 23, CDR-L2 с SEQ ID NO: 24 и CDR-L3 с SEQ ID NO: 25.
В одном аспекте антигенсвязывающий домен, способный к специфическому связыванию с CD28, биспецифической агонистической CD28-антигенсвязывающей молекулы содержит вариабельную область тяжелой цепи (VHCD28), содержащую CDR-H1 с SEQ ID NO: 36, CDR-H2 с SEQ ID NO: 37 и CDR-H3 с SEQ ID NO: 38, и вариабельную область легкой цепи (VLCD28), содержащую CDR-L1 с SEQ ID NO: 39, CDR-L2 с SEQ ID NO: 40 и CDR-L3 с SEQ ID NO: 41.
В другом аспекте антигенсвязывающий домен, способный к специфическому связыванию с CD28, биспецифической агонистической CD28-антигенсвязывающей молекулы содержит вариабельную область тяжелой цепи (VHCD28), содержащую CDR-H1 с SEQ ID NO: 20, CDR-H2 с SEQ ID NO: 21 и CDR-H3 с SEQ ID NO: 22, и вариабельную область легкой цепи (VLCD28), содержащую CDR-L1 с SEQ ID NO: 23, CDR-L2 с SEQ ID NO: 24 и CDR-L3 с SEQ ID NO: 25.
Кроме того, предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула по определению выше в данном документе, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD28, содержит вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO:26, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO:27.
В дополнительном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD28, содержит вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO:42, SEQ ID NO:43, SEQ ID NO:44, SEQ ID NO:45, SEQ ID NO:46, SEQ ID NO:47, SEQ ID NO:48, SEQ ID NO:49, SEQ ID NO:50 и SEQ ID NO:51, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO:27, SEQ ID NO:52, SEQ ID NO:53, SEQ ID NO:54, SEQ ID NO:55, SEQ ID NO:56, SEQ ID NO:57, SEQ ID NO:58, SEQ ID NO:59, SEQ ID NO:60 и SEQ ID NO:61.
В другом аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD28, содержит
(а) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO:47, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO:54, или
(б) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO:47, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO:27, или
(в) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO:51, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO:61, или
(г) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO:46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO:53, или
(д) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO:46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO:54, или
(е) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO:46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO:59, или
(ж) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO:46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO:27, или
(и) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO:43, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO:27, или
(к) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO:42, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO:53, или
(л) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO:42, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO:59, или
(м) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO:42, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO:27.
В одном конкретном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD28, содержит вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO:47, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO:54.
В другом конкретном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD28, содержит вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO:46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO:53.
В дополнительном конкретном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD28, содержит вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO:42, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO:27.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с опухолеассоциированным антигеном, представляет собой антигенсвязывающий домен, способный к специфическому связыванию с карциноэмбриональным антигеном (СЕА).
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, в которой по меньшей мере один антигенсвязывающий домен, способный к специфическому связыванию с СЕА, содержит
(i) вариабельную область тяжелой цепи (VHCEA), содержащую CDR-H1, содержащую аминокислотную последовательность SEQ ID NO: 188, CDR-H2, содержащую аминокислотную последовательность SEQ ID NO: 189, и CDR-H3, содержащую аминокислотную последовательность SEQ ID NO:190, и вариабельную область легкой цепи (VLCEA), содержащую CDR-L1, содержащую аминокислотную последовательность SEQ ID NO: 191, CDR-L2, содержащую аминокислотную последовательность SEQ ID NO: 192, и CDR-L3, содержащую аминокислотную последовательность SEQ ID NO: 193; или
(ii) вариабельную область тяжелой цепи (VHCEA), содержащую CDR-H1, содержащую аминокислотную последовательность SEQ ID NO:180, CDR-H2, содержащую аминокислотную последовательность SEQ ID NO: 181, и CDR-H3, содержащую аминокислотную последовательность SEQ ID NO: 182, и вариабельную область легкой цепи (VLCEA), содержащую CDR-L1, содержащую аминокислотную последовательность SEQ ID NO: 183, CDR-L2, содержащую аминокислотную последовательность SEQ ID NO: 184, и CDR-L3, содержащую аминокислотную последовательность SEQ ID NO: 185; или
(iii) вариабельную область тяжелой цепи (VHCEA), содержащую CDR-H1, содержащую аминокислотную последовательность SEQ ID NO: 127, CDR-H2, содержащую аминокислотную последовательность SEQ ID NO: 128, и CDR-H3, содержащую аминокислотную последовательность SEQ ID NO: 129, и вариабельную область легкой цепи (VLCEA), содержащую CDR-L1, содержащую аминокислотную последовательность SEQ ID NO:130, CDR-L2, содержащую аминокислотную последовательность SEQ ID NO: 131, и CDR-L3, содержащую аминокислотную последовательность SEQ ID NO: 132, или
(iv) вариабельную область тяжелой цепи (VHCEA), содержащую CDR-H1, содержащую аминокислотную последовательность SEQ ID NO: 507, CDR-H2, содержащую аминокислотную последовательность SEQ ID NO: 508, и CDR-H3, содержащую аминокислотную последовательность SEQ ID NO: 509, и вариабельную область легкой цепи (VLCEA), содержащую CDR-L1, содержащую аминокислотную последовательность SEQ ID NO: 510, CDR-L2, содержащую аминокислотную последовательность SEQ ID NO: 511, и CDR-L3, содержащую аминокислотную последовательность SEQ ID NO: 512.
В одном аспекте антигенсвязывающий домен, способный к специфическому связыванию с СЕА, содержит вариабельную область тяжелой цепи (VHCEA), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 133, и вариабельную область легкой цепи (VLCEA), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 134. В частности, антигенсвязывающий домен, способный к специфическому связыванию с СЕА, содержит вариабельную область тяжелой цепи (VHCEA), содержащую аминокислотную последовательность SEQ ID NO: 186, и вариабельную область легкой цепи (VLCEA), содержащую аминокислотную последовательность SEQ ID NO: 187.
В другом аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с СЕА, содержит
(а) вариабельную область тяжелой цепи (VHCEA), содержащую аминокислотную последовательность SEQ ID NO: 194, и вариабельную область легкой цепи (VLCEA), содержащую аминокислотную последовательность SEQ ID NO:195, или
(б) вариабельную область тяжелой цепи (VHCEA), содержащую аминокислотную последовательность SEQ ID NO: 196, и вариабельную область легкой цепи (VLCEA), содержащую аминокислотную последовательность SEQ ID NO: 197, или
(в) вариабельную область тяжелой цепи (VHCEA), содержащую аминокислотную последовательность SEQ ID NO: 198, и вариабельную область легкой цепи (VLCEA), содержащую аминокислотную последовательность SEQ ID NO: 199, или
(г) вариабельную область тяжелой цепи (VHCEA), содержащую аминокислотную последовательность SEQ ID NO: 200, и вариабельную область легкой цепи (VLCEA), содержащую аминокислотную последовательность SEQ ID NO: 201, или
(д) вариабельную область тяжелой цепи (VHCEA), содержащую аминокислотную последовательность SEQ ID NO: 202, и вариабельную область легкой цепи (VLCEA), содержащую аминокислотную последовательность SEQ ID NO: 203, или
(е) вариабельную область тяжелой цепи (VHCEA), содержащую аминокислотную последовательность SEQ ID NO: 204, и вариабельную область легкой цепи (VLCEA), содержащую аминокислотную последовательность SEQ ID NO: 205, или
(ж) вариабельную область тяжелой цепи (VHCEA), содержащую аминокислотную последовательность SEQ ID NO: 206, и вариабельную область легкой цепи (VLCEA), содержащую аминокислотную последовательность SEQ ID NO: 207, или
(и) вариабельную область тяжелой цепи (VHCEA), содержащую аминокислотную последовательность SEQ ID NO: 208, и вариабельную область легкой цепи (VLCEA), содержащую аминокислотную последовательность SEQ ID NO: 209, или
(к) вариабельную область тяжелой цепи (VHCEA), содержащую аминокислотную последовательность SEQ ID NO: 210, и вариабельную область легкой цепи (VLCEA), содержащую аминокислотную последовательность SEQ ID NO: 211, или
(л) вариабельную область тяжелой цепи (VHCEA), содержащую аминокислотную последовательность SEQ ID NO: 212, и вариабельную область легкой цепи (VLCEA), содержащую аминокислотную последовательность SEQ ID NO: 213.
В частности, антигенсвязывающий домен, способный к специфическому связыванию с СЕА, содержит вариабельную область тяжелой цепи (VHCEA), содержащую аминокислотную последовательность SEQ ID NO: 200, и вариабельную область легкой цепи (VLCEA), содержащую аминокислотную последовательность SEQ ID NO: 201.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с опухолеассоциированным антигеном, представляет собой антигенсвязывающий домен, способный к специфическому связыванию с белком активации фибробластов (FAP). В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, в которой антигенсвязывающий домен, способный к специфическому связыванию с FAP, содержит (а) вариабельную область тяжелой цепи (VHFAP), содержащую (i) CDR-H1, содержащую аминокислотную последовательность SEQ ID NO: 12, (ii) CDR-H2, содержащую аминокислотную последовательность SEQ ID NO: 13, и (iii) CDR-H3, содержащую аминокислотную последовательность SEQ ID NO: 14, и вариабельную область легкой цепи (VLFAP), содержащую (iv) CDR-L1, содержащую аминокислотную последовательность SEQ ID NO: 15, (v) CDR-L2, содержащую аминокислотную последовательность SEQ ID NO: 16, и (vi) CDR-L3, содержащую аминокислотную последовательность SEQ ID NO: 17, или (б) вариабельную область тяжелой цепи (VHFAP), содержащую (i) CDR-H1, содержащую аминокислотную последовательность SEQ ID NO: 4, (ii) CDR-H2, содержащую аминокислотную последовательность SEQ ID NO: 5, и (iii) CDR-H3, содержащую аминокислотную последовательность SEQ ID NO: 6, и вариабельную область легкой цепи (VLFAP), содержащую (iv) CDR-L1, содержащую аминокислотную последовательность SEQ ID NO: 7, (v) CDR-L2, содержащую аминокислотную последовательность SEQ ID NO: 8, и (vi) CDR-L3, содержащую аминокислотную последовательность SEQ ID NO: 9. В частности, антигенсвязывающий домен, способный к специфическому связыванию с FAP, содержит вариабельную область тяжелой цепи (VHFAP), содержащую (i) CDR-Н1, содержащую аминокислотную последовательность SEQ ID NO: 12, (ii) CDR-H2, содержащую аминокислотную последовательность SEQ ID NO: 13, и (iii) CDR-H3, содержащую аминокислотную последовательность SEQ ID NO: 14, и вариабельную область легкой цепи (VLFAP), содержащую (iv) CDR-L1, содержащую аминокислотную последовательность SEQ ID NO: 15, (v) CDR-L2, содержащую аминокислотную последовательность SEQ ID NO: 16, и (vi) CDR-L3, содержащую аминокислотную последовательность SEQ ID NO: 17. В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с FAP, содержит (а) вариабельную область тяжелой цепи (VHFAP), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 18, и вариабельную область легкой цепи (VLFAP), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 19, или (б) вариабельную область тяжелой цепи (VHFAP), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 10, и вариабельную область легкой цепи (VLFAP), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 11. В частности, антигенсвязывающий домен, способный к специфическому связыванию с FAP, содержит вариабельную область тяжелой цепи (VHFAP), содержащую аминокислотную последовательность SEQ ID NO: 18, и вариабельную область легкой цепи (VLFAP), содержащую аминокислотную последовательность SEQ ID NO: 19.
В другом аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с опухолеассоциированным антигеном, представляет собой антигенсвязывающий домен, способный к специфическому связыванию с молекулой адгезии эпителиальных клеток (ЕрСАМ). В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, в которой антигенсвязывающий домен, способный к специфическому связыванию с ЕрСАМ, содержит вариабельную область тяжелой цепи (VHEpCAM), содержащую (i) CDR-H1, содержащую аминокислотную последовательность SEQ ID NO: 515, (ii) CDR-H2, содержащую аминокислотную последовательность SEQ ID NO: 516, и (iii) CDR-H3, содержащую аминокислотную последовательность SEQ ID NO: 517, и вариабельную область легкой цепи (VLEpCAM), содержащую (iv) CDR-L1, содержащую аминокислотную последовательность SEQ ID NO: 518, (v) CDR-L2, содержащую аминокислотную последовательность SEQ ID NO: 519, и (vi) CDR-L3, содержащую аминокислотную последовательность SEQ ID NO: 520. В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с ЕрСАМ, содержит (а) вариабельную область тяжелой цепи (VHEpCAM), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 521, и вариабельную область легкой цепи (VLEpCAM), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 522. В частности, антигенсвязывающий домен, способный к специфическому связыванию с ЕрСАМ, содержит вариабельную область тяжелой цепи (VHEpCAM), содержащую аминокислотную последовательность SEQ ID NO: 521, и вариабельную область легкой цепи (VLEpCAM), содержащую аминокислотную последовательность SEQ ID NO: 522.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с опухолеассоциированным антигеном, представляет собой антигенсвязывающий домен, способный к специфическому связыванию с HER3. В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, в которой антигенсвязывающий домен, способный к специфическому связыванию с HER3, содержит вариабельную область тяжелой цепи (VHHER3), содержащую (i) CDR-H1, содержащую аминокислотную последовательность SEQ ID NO: 523, (ii) CDR-H2, содержащую аминокислотную последовательность SEQ ID NO: 524, и (iii) CDR-H3, содержащую аминокислотную последовательность SEQ ID NO: 525, и вариабельную область легкой цепи (VLHER3), содержащую (iv) CDR-L1, содержащую аминокислотную последовательность SEQ ID NO: 526, (v) CDR-L2, содержащую аминокислотную последовательность SEQ ID NO: 527, и (vi) CDR-L3, содержащую аминокислотную последовательность SEQ ID NO: 528. В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с HER3, содержит (а) вариабельную область тяжелой цепи (VHHER3), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 529, и вариабельную область легкой цепи (VLHER3), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 530. В частности, антигенсвязывающий домен, способный к специфическому связыванию с HER3, содержит вариабельную область тяжелой цепи (VHHER3), содержащую аминокислотную последовательность SEQ ID NO: 529, и вариабельную область легкой цепи (VLHER3), содержащую аминокислотную последовательность SEQ ID NO: 530.
В еще одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с опухолеассоциированным антигеном, представляет собой антигенсвязывающий домен, способный к специфическому связыванию с CD30. В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD30, содержит вариабельную область тяжелой цепи (VHCD30), содержащую (i) CDR-H1, содержащую аминокислотную последовательность SEQ ID NO: 531, (ii) CDR-H2, содержащую аминокислотную последовательность SEQ ID NO: 532, и (iii) CDR-H3, содержащую аминокислотную последовательность SEQ ID NO: 533, и вариабельную область легкой цепи (VLCD30), содержащую (iv) CDR-L1, содержащую аминокислотную последовательность SEQ ID NO: 534, (v) CDR-L2, содержащую аминокислотную последовательность SEQ ID NO: 535, и (vi) CDR-L3, содержащую аминокислотную последовательность SEQ ID NO: 536. В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD30, содержит (а) вариабельную область тяжелой цепи (VHCD30), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 537, и вариабельную область легкой цепи (VLCD30), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 538. В частности, антигенсвязывающий домен, способный к специфическому связыванию с CD30, содержит вариабельную область тяжелой цепи (VHCD30), содержащую аминокислотную последовательность SEQ ID NO: 537, и вариабельную область легкой цепи (VLCD30), содержащую аминокислотную последовательность SEQ ID NO: 538.
Кроме того, предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с опухолеассоциированным антигеном, представляет собой антигенсвязывающий домен, способный к специфическому связыванию с TBPG. В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, в которой антигенсвязывающий домен, способный к специфическому связыванию с TBPG, содержит вариабельную область тяжелой цепи (VHTBPG), содержащую (i) CDR-H1, содержащую аминокислотную последовательность SEQ ID NO: 539, (ii) CDR-H2, содержащую аминокислотную последовательность SEQ ID NO: 540, и (iii) CDR-H3, содержащую аминокислотную последовательность SEQ ID NO: 541, и вариабельную область легкой цепи (VLTBPG), содержащую (iv) CDR-L1, содержащую аминокислотную последовательность SEQ ID NO: 542, (v) CDR-L2, содержащую аминокислотную последовательность SEQ ID NO: 543, и (vi) CDR-L3, содержащую аминокислотную последовательность SEQ ID NO: 544. В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с TBPG, содержит (а) вариабельную область тяжелой цепи (VHTBPG), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 545, и вариабельную область легкой цепи (VLTBPG), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 546. В частности, антигенсвязывающий домен, способный к специфическому связыванию с TBPG, содержит вариабельную область тяжелой цепи (VHTBPG), содержащую аминокислотную последовательность SEQ ID NO: 545, и вариабельную область легкой цепи (VLTBPG), содержащую аминокислотную последовательность SEQ ID NO: 546.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, характеризующаяся одновалентным связыванием с CD28, содержащая (а) один антигенсвязывающий домен, способный к специфическому связыванию с CD28, (б) по меньшей мере один антигенсвязывающий домен, способный к специфическому связыванию с антигеном клеточной поверхности множественной миеломы (ММ), и (в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию. В одном аспекте антиген клеточной поверхности множественной миеломы (ММ) выбран из группы, состоящей из CD38, ВСМА и GPRC5D.
Таким образом, предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с опухолеассоциированным антигеном, представляет собой антигенсвязывающий домен, способный к специфическому связыванию с GPRC5D. В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, в которой антигенсвязывающий домен, способный к специфическому связыванию с GPRC5D, содержит (а) вариабельную область тяжелой цепи (VHGPRC5D), содержащую (i) CDR-H1, содержащую аминокислотную последовательность SEQ ID NO: 563, (ii) CDR-H2, содержащую аминокислотную последовательность SEQ ID NO: 564, и (iii) CDR-Н3, содержащую аминокислотную последовательность SEQ ID NO: 565, и вариабельную область легкой цепи (VLGPRC5D), содержащую (iv) CDR-L1, содержащую аминокислотную последовательность SEQ ID NO: 566, (v) CDR-L2, содержащую аминокислотную последовательность SEQ ID NO: 567, и (vi) CDR-L3, содержащую аминокислотную последовательность SEQ ID NO: 568, или (б) вариабельную область тяжелой цепи (VHGPRC5D), содержащую (i) CDR-H1, содержащую аминокислотную последовательность SEQ ID NO: 579, (ii) CDR-H2, содержащую аминокислотную последовательность SEQ ID NO: 580, и (iii) CDR-Н3, содержащую аминокислотную последовательность SEQ ID NO: 581, и вариабельную область легкой цепи (VLGPRC5D), содержащую (iv) CDR-L1, содержащую аминокислотную последовательность SEQ ID NO: 582, (v) CDR-L2, содержащую аминокислотную последовательность SEQ ID NO: 583, и (vi) CDR-L3, содержащую аминокислотную последовательность SEQ ID NO: 584. В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с GPRC5D, содержит вариабельную область тяжелой цепи (VHGPRC5D), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 569, и вариабельную область легкой цепи (VLGPRC5D), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 570. В частности, антигенсвязывающий домен, способный к специфическому связыванию с GPRC5D, содержит вариабельную область тяжелой цепи (VHGPRC5D), содержащую аминокислотную последовательность SEQ ID NO: 569, и вариабельную область легкой цепи (VLGPRC5D), содержащую аминокислотную последовательность SEQ ID NO: 570.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с опухолеассоциированным антигеном, представляет собой антигенсвязывающий домен, способный к специфическому связыванию с CD38. В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD38, содержит вариабельную область тяжелой цепи (VHCD38), содержащую (i) CDR-H1, содержащую аминокислотную последовательность SEQ ID NO: 547, (ii) CDR-H2, содержащую аминокислотную последовательность SEQ ID NO: 548, и (iii) CDR-H3, содержащую аминокислотную последовательность SEQ ID NO: 549, и вариабельную область легкой цепи (VLCD38), содержащую (iv) CDR-L1, содержащую аминокислотную последовательность SEQ ID NO: 550, (v) CDR-L2, содержащую аминокислотную последовательность SEQ ID NO: 551, и (vi) CDR-L3, содержащую аминокислотную последовательность SEQ ID NO: 552. В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD38, содержит вариабельную область тяжелой цепи (VHCD38), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 553, и вариабельную область легкой цепи (VLCD38), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 554. В частности, антигенсвязывающий домен, способный к специфическому связыванию с CD38, содержит вариабельную область тяжелой цепи (VHCD38), содержащую аминокислотную последовательность SEQ ID NO: 553, и вариабельную область легкой цепи (VLCD38), содержащую аминокислотную последовательность SEQ ID NO: 554.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с опухолеассоциированным антигеном, представляет собой антигенсвязывающий домен, способный к специфическому связыванию с ВСМА. В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, в которой антигенсвязывающий домен, способный к специфическому связыванию с ВСМА, содержит вариабельную область тяжелой цепи (VHBCMA), содержащую (i) CDR-H1, содержащую аминокислотную последовательность SEQ ID NO: 555, (ii) CDR-H2, содержащую аминокислотную последовательность SEQ ID NO: 556, и (iii) CDR-H3, содержащую аминокислотную последовательность SEQ ID NO: 557, и вариабельную область легкой цепи (VLBCMA), содержащую (iv) CDR-L1, содержащую аминокислотную последовательность SEQ ID NO: 558, (v) CDR-L2, содержащую аминокислотную последовательность SEQ ID NO: 559, и (vi) CDR-L3, содержащую аминокислотную последовательность SEQ ID NO: 560. В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с ВСМА, содержит вариабельную область тяжелой цепи (VHBCMA), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 559, и вариабельную область легкой цепи (VLBCMA), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 560. В частности, антигенсвязывающий домен, способный к специфическому связыванию с ВСМА, содержит вариабельную область тяжелой цепи (VHBCMA), содержащую аминокислотную последовательность SEQ ID NO: 561, и вариабельную область легкой цепи (VLBCMA), содержащую аминокислотную последовательность SEQ ID NO: 562.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, характеризующаяся одновалентным связыванием с CD28, содержащая (а) один антигенсвязывающий домен, способный к специфическому связыванию с CD28, (б) по меньшей мере один антигенсвязывающий домен, способный к специфическому связыванию с поверхностным антигеном В-клеток, и (в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию. В одном аспекте поверхностный антиген В-клеток выбран из группы, состоящей из CD19, CD79b, CD20, CD22 и CD37.
Таким образом, предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с опухолеассоциированным антигеном, представляет собой антигенсвязывающий домен, способный к специфическому связыванию с CD19. В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD19, содержит (а) вариабельную область тяжелой цепи (VHCD19), содержащую (i) CDR-H1, содержащую аминокислотную последовательность SEQ ID NO: 406, (ii) CDR-H2, содержащую аминокислотную последовательность SEQ ID NO: 407, и (iii) CDR-H3, содержащую аминокислотную последовательность SEQ ID NO: 408, и вариабельную область легкой цепи (VLCD19), содержащую (iv) CDR-L1, содержащую аминокислотную последовательность SEQ ID NO:409, (v) CDR-L2, содержащую аминокислотную последовательность SEQ ID NO:410, и (vi) CDR-L3, содержащую аминокислотную последовательность SEQ ID NO: 411, или (б) вариабельную область тяжелой цепи (VHCD19), содержащую (i) CDR-H1, содержащую аминокислотную последовательность SEQ ID NO: 414, (ii) CDR-H2, содержащую аминокислотную последовательность SEQ ID NO: 415, и (iii) CDR-Н3, содержащую аминокислотную последовательность SEQ ID NO: 416, и вариабельную область легкой цепи (VLCD19), содержащую (iv) CDR-L1, содержащую аминокислотную последовательность SEQ ID NO: 417, (v) CDR-L2, содержащую аминокислотную последовательность SEQ ID NO: 418, и (vi) CDR-L3, содержащую аминокислотную последовательность SEQ ID NO: 419. В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD19, содержит (а) вариабельную область тяжелой цепи (VHCD19), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 98% или 100% идентичной аминокислотной последовательности SEQ ID NO: 412, и вариабельную область легкой цепи (VLCD19), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 98% или 100% идентичной аминокислотной последовательности SEQ ID NO: 413, или (б) вариабельную область тяжелой цепи (VHCD19), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 98% или 100% идентичной аминокислотной последовательности SEQ ID NO: 420, и вариабельную область легкой цепи (VLCD19), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 98% или 100% идентичной аминокислотной последовательности SEQ ID ΝΟ: 421. В частности, антигенсвязывающий домен, способный к специфическому связыванию с CD19, содержит вариабельную область тяжелой цепи (VHCD19), содержащую аминокислотную последовательность SEQ ID NO: 412, и вариабельную область легкой цепи (VLCD19), содержащую аминокислотную последовательность SEQ ID NO: 413.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с опухолеассоциированным антигеном, представляет собой антигенсвязывающий домен, способный к специфическому связыванию с CD79b. В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD79b, содержит вариабельную область тяжелой цепи (VHCD79b), содержащую (i) CDR-H1, содержащую аминокислотную последовательность SEQ ID NO: 422, (ii) CDR-H2, содержащую аминокислотную последовательность SEQ ID NO: 423, и (iii) CDR-H3, содержащую аминокислотную последовательность SEQ ID NO: 424, и вариабельную область легкой цепи (VLCD79b), содержащую (iv) CDR-L1, содержащую аминокислотную последовательность SEQ ID NO: 425, (v) CDR-L2, содержащую аминокислотную последовательность SEQ ID NO: 426, и (vi) CDR-L3, содержащую аминокислотную последовательность SEQ ID NO: 427. В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD79b, содержит вариабельную область тяжелой цепи (VHCD79b), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 428, и вариабельную область легкой цепи (VLCD79b), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 429. В частности, антигенсвязывающий домен, способный к специфическому связыванию с CD79b, содержит вариабельную область тяжелой цепи (VHCD79b), содержащую аминокислотную последовательность SEQ ID NO: 428, и вариабельную область легкой цепи (VLCD79b), содержащую аминокислотную последовательность SEQ ID NO: 429.
В дополнительном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула по определению выше в данном документе, в которой по меньшей мере один антигенсвязывающий домен, способный к специфическому связыванию с CD28, представляет собой Fab-фрагмент или кросс-Fab-фрагмент.
В другом аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один Fab-фрагмент, способный к специфическому связыванию с CD28,
(б) один kpocc-Fab-фрагмент, способный к специфическому связыванию с опухолеассоциированным антигеном, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В дополнительном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) первый Fab-фрагмент, способный к специфическому связыванию с CD28,
(б) второй Fab-фрагмент, способный к специфическому связыванию с опухолеассоциированным антигеном, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию,
причем первый Fab-фрагмент, способный к специфическому связыванию с CD28, слит на С-конце тяжелой цепи Fab с Ν-концом тяжелой цепи Fab второго Fab-фрагмента, способного к специфическому связыванию с опухолеассоциированным антигеном, который в свою очередь слит на своем С-конце с Ν-концом одной из субъединиц Fc-домена.
В другом аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) первый Fab-фрагмент, способный к специфическому связыванию с CD28,
(б) второй и третий Fab-фрагменты, способные к специфическому связыванию с опухолеассоциированным антигеном, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию,
причем первый Fab-фрагмент, способный к специфическому связыванию с CD28, слит на С-конце тяжелой цепи Fab с Ν-концом тяжелой цепи Fab второго Fab-фрагмента, способного к специфическому связыванию с опухолеассоциированным антигеном, который в свою очередь слит на своем С-конце с Ν-концом первой субъединицы Fc-домена, и третий Fab-фрагмент, способный к специфическому связыванию с опухолеассоциированным антигеном, слит на С-конце тяжелой цепи Fab с Ν-концом второй субъединицы Fc-домена.
В дополнительном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) Fab-фрагмент, способный к специфическому связыванию с CD28,
(б) домены VH и VL, способные к специфическому связыванию с опухолеассоциированным антигеном, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию,
причем Fab-фрагмент, способный к специфическому связыванию с CD28, слит на своем С-конце с Ν-концом первой субъединицы Fc-домена, и при этом один из доменов VH и VL, способных к специфическому связыванию с опухолеассоциированным антигеном, слит посредством пептидного линкера с С-концом первой субъединицы Fc-домена, а другой из доменов VH и VL, способных к специфическому связыванию с опухолеассоциированным антигеном, слит посредством пептидного линкера с С-концом второй субъединицы Fc-домена.
В соответствии с другим аспектом изобретения предложены один или более выделенных полинуклеотидов, кодирующих биспецифическую агонистическую CD28-антигенсвязывающую молекулу по изобретению. В изобретении дополнительно предложены один или более векторов, в частности, экспрессионных векторов, содержащих выделенные полинуклеотиды по изобретению, и клетка-хозяин, содержащая выделенные полинуклеотиды или экспрессионные векторы по изобретению. В некоторых аспектах клетка-хозяин представляет собой эукариотическую клетку, в частности, клетку млекопитающего. В другом аспекте предложен способ получения биспецифической агонистической CD28-антигенсвязывающей молекулы, описанной в данном документе, включающий культивирование клетки-хозяина по изобретению в условиях, подходящих для экспрессии биспецифической агонистической CD28-антигенсвязывающей молекулы. Необязательно, способ также включает выделение биспецифической агонистической CD28-антигенсвязывающей молекулы. Изобретение также включает биспецифическую агонистическую CD28-антигенсвязывающую молекулу, полученную способом по изобретению.
В изобретении дополнительно предложена фармацевтическая композиция, содержащая биспецифическую агонистическую CD28-антигенсвязывающую молекулу по изобретению и по меньшей мере один фармацевтически приемлемый эксципиент.В одном аспекте фармацевтическая композиция предназначена для применения в лечении рака.
Также изобретение охватывает способы применения биспецифической агонистической CD28-антигенсвязывающей молекулы и фармацевтической композиции по изобретению. В одном аспекте в изобретении предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула или фармацевтическая композиция в соответствии с изобретением для применения в качестве лекарственного средства. В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, для применения в (а) повышении активации или (б) повышении эффекторных функций Т-клеток. В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула или фармацевтическая композиция в соответствии с изобретением для применения в лечении заболевания. В конкретном аспекте заболевание представляет собой рак. В другом аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула или фармацевтическая композиция в соответствии с изобретением для применения в лечении рака, причем биспецифическую агонистическую CD28-антигенсвязывающую молекулу вводят в комбинации с химиотерапевтическим агентом, лучевой терапией и/или другими агентами для применения в иммунотерапии рака. В дополнительном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула или фармацевтическая композиция в соответствии с изобретением для применения в лечении рака, причем биспецифическую агонистическую CD28-антигенсвязывающую молекулу вводят в комбинации с активирующим Т-клетки биспецифическим анти-CD3 антителом. В дополнительном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула или фармацевтическая композиция в соответствии с изобретением для применения в лечении рака, причем биспецифическую агонистическую CD28-антигенсвязывающую молекулу вводят в комбинации с анти-PD-L1 антителом или анти-PD-1 антителом.
Также предложено применение биспецифической агонистической CD28-антигенсвязывающей молекулы в соответствии с изобретением в производстве лекарственного средства для лечения рака; а также способ лечения заболевания у субъекта, включающий введение указанному субъекту терапевтически эффективного количества биспецифической агонистической CD28-антигенсвязывающей молекулы в соответствии с изобретением или композиции, содержащей биспецифическую агонистическую CD28-антигенсвязывающую молекулу в соответствии с изобретением в фармацевтически приемлемой форме. В конкретном аспекте заболевание представляет собой рак. В одном аспекте предложен способ (а) повышения активации или (б) повышения эффекторных функций Т-клеток у субъекта, включающий введение указанному субъекту биспецифической агонистической CD28-антигенсвязывающей молекулы в соответствии с изобретением или композиции, содержащей биспецифическую агонистическую CD28-антигенсвязывающую молекулу в соответствии с изобретением в фармацевтически приемлемой форме. В другом аспекте предложено применение биспецифической агонистической CD28-антигенсвязывающей молекулы в соответствии с изобретением в производстве лекарственного средства для лечения заболевания, причем лечение включает совместное введение с химиотерапевтическим агентом, лучевой терапией и/или другими агентами для применения в иммунотерапии рака. В дополнительном аспекте предложен способ лечения заболевания у субъекта, включающий введение указанному субъекту терапевтически эффективного количества биспецифической агонистической CD28-антигенсвязывающей молекулы в соответствии с изобретением или композиции, содержащей биспецифическую агонистическую CD28-антигенсвязывающую молекулу в соответствии с изобретением в фармацевтически приемлемой форме, причем способ включает совместное введение с химиотерапевтическим агентом, лучевой терапией и/или другими агентами для применения в иммунотерапии рака. В дополнительном аспекте предложен способ лечения заболевания у субъекта, включающий введение указанному субъекту терапевтически эффективного количества биспецифической агонистической CD28-антигенсвязывающей молекулы в соответствии с изобретением или композиции, содержащей биспецифическую агонистическую CD28-антигенсвязывающую молекулу в соответствии с изобретением в фармацевтически приемлемой форме, причем способ включает совместное введение с активирующим Т-клетки биспецифическим анти-CD3 антителом. В другом аспекте предложен способ лечения заболевания у субъекта, включающий введение указанному субъекту терапевтически эффективного количества биспецифической агонистической CD28-антигенсвязывающей молекулы в соответствии с изобретением или композиции, содержащей биспецифическую агонистическую CD28-антигенсвязывающую молекулу в соответствии с изобретением в фармацевтически приемлемой форме, причем способ включает совместное введение с анти-PD-L1 антителом или анти-PD-1 антителом. Также предложен способ ингибирования роста опухолевых клеток у субъекта, включающий введение субъекту эффективного количества биспецифической агонистической CD28-антигенсвязывающей молекулы в соответствии с изобретением или композиции, содержащей биспецифическую агонистическую CD28-антигенсвязывающую молекулу в соответствии с изобретением в фармацевтически приемлемой форме, для ингибирования роста опухолевых клеток. В любом из вышеприведенных аспектов субъект предпочтительно представляет собой млекопитающее, в частности, человека.
КРАТКОЕ ОПИСАНИЕ ГРАФИЧЕСКИХ МАТЕРИАЛОВ
На Фиг. 1А-1Н приведены схематические иллюстрации описанных молекул. На Фиг. 1А показано агонистическое антитело к CD28 - CD28(SA) в изоформе huIgG4 (TGN1412).
На Фиг. 1Б проиллюстрировано агонистическое антитело CD28(SA) в виде изотипа hu IgG1 PGLALA («Fc-сайленсинг»).
Биспецифические антигенсвязывающие молекулы FAP-CD28 в формате 1+1, формате 1+2, формате 2+2 и формате 1+4 показаны на Фиг. 1 В, 1Г, 1Д и 1Е, соответственно.
Биспецифические антигенсвязывающие молекулы CEA-CD28 в формате 1+2, формате 2+2 и формате 1+1 показаны на Фиг. 1Ж, 1И и 1Л, соответственно.
На Фиг. 1К приведена схематическая иллюстрация вариантов агонистического антитела к CD28 в виде одновалентного изотипа hu IgG1 PGLALA («Fc-сайленсинг»).
На Фиг. 1М показана биспецифическая антигенсвязывающая молекула FAP-CD28 в формате 1+1, в которой FAP-антигенсвязывающий домен представлен доменами VH и VL, каждый из которых слит с одним С-концом субъединиц Fc-домена.
На Фиг. 1Н проиллюстрирована биспецифическая антигенсвязывающая молекула FAP-CD28 в формате 2+1, в которой CD28-антигенсвязывающий домен представлен кросс-Fab, который слит в своем С-конце с Ν-концом другой тяжелой цепи «двухвалентного» антитела к FAP.
На Фиг. 1П показана другая биспецифическая антигенсвязывающая молекула FAP-CD28 в формате 1+1, в которой CD28-антигенсвязывающий домен представлен кросс-Fab, который слит в своем С-конце с Ν-концом Fab-фрагмента, связывающегося с FAP.
На Фиг. 1Р проиллюстрирована триспецифическая антигенсвязывающая молекула FAP-CEA-CD28 в формате 1+1+1, в которой CD28-антигенсвязывающий домен представлен Fab, который слит в С-конце как легкой, так и тяжелой цепи с Ν-концом как легкой, так и тяжелой цепи анти-FAP антигенсвязывающего домена на цепи Fc-выступа hulgG1 PG-LALA, и в которой анти-СЕА кросс-Fab фрагмент является частью цепи Fc-впадины huIgG1 PG-LALA.
Фиг. 2А, 2Б, 2В, 2Г и 2Д относятся к связыванию агонистических антител к CD28 и антигенсвязывающих молекул FAP-CD28 с CD28 человека или FAP человека на клетках. Показано связывание CD28(SA) в изоформе IgG4 в сравнении с изотипом hu IgG1 PGLALA против CD28 человека на Фиг. 2А и связывание разных молекул FAP-CD28 с CD28 человека (Фиг. 2Б) и FAP человека (Фиг. 2В) на клетках. Медианную интенсивность флуоресценции для связывания разных агонистических антител к CD28 или анти-DP47 нацеленных молекул с клетками СНО, экспрессирующими CD28 человека (родительская линия клеток CHO-k1 АТСС №CCL-61, модифицированная для стабильной сверхэкспрессии CD28 человека), или клетками 3Т3, экспрессирующими FAP человека (линия клеток NIH/3T3 (АТСС CRL-1658)), оценивали методом проточной цитометрии. Изображены три технических повтора с СПС. Сравнение антигенсвязывающих молекул FAP(4B9)-CD28(SA) (молекулы D, Ε и F, описанные в примере 1) приведено на Фиг. 2Г (связывание с CD28 человека) и Фиг. 2Д (связывание с FAP человека).
На Фиг. 2Е и 2Ж показано связывание разных форматов антигенсвязывающих молекул FAP-CD28 с одновалентным связыванием с CD28 и FAP, соответственно. Показаны кривые для FAP-CD28 CTF 1+1 (Р1АЕ2236, молекула I), FAP-CD28 1+1 (P1AD4492, молекула С), FAP-CD28 Н2Т 1+1 (Р1АЕ2021, молекула Н) и двух соединений FAP-CD28(SA) 1+2 (P1AD9011, молекула Е) и DP47 в качестве референса. Приведена медианная интенсивность флуоресценции для связывания антител FAP-CD28 или анти-DP47 антитела (отрицательный контроль) с клетками СНО, экспрессирующими CD28 человека (родительская линия клеток CHO-k1 АТСС № CCL-61, модифицированная для стабильной сверхэкспрессии CD28 человека) (Фиг. 2Е), или клетками 3Т3, экспрессирующими FAP человека (линия клеток NIH/3T3 (АТСС CRL-1658)) (Фиг. 2Ж), которую оценивали методом проточной цитометрии. Приведены три технических повтора с СПС. На Фиг. 2И и 2К показано связывание FAP-CD28 2+1 (Р1АЕ5231, молекула Ж) с CD28 и FAP, соответственно.
Выравнивание вариабельных доменов CD28(SA) и их вариантов показано на Фиг. 3А-3Г. Выравнивание домена VH CD28(SA) и его вариантов с целью удаления цистеина 50 и снижения в разной степени аффинности получаемых в результате анти-CD28 связывающих молекул показано на Фиг. 3А и 3Б. Следует отметить, что в вариантах VH к и л CDR CD28(SA) прививали из каркасной области IGHV1-2 в каркасную область IGHV3-23 (Фиг. 3Б). На Фиг. 3В и 3Г показано выравнивание домена VL CD28(SA) и его вариантов с целью снижения в разной степени аффинности получаемых в результате анти-CD28 связывающих молекул. В варианте ц CDR прививали в каркасную последовательность VL-последовательности трастузумаба (Герцептин).
На Фиг. 4А-4Б показано связывание вариантов агонистического антитела к CD28 со сниженной аффинностью в моноспецифических одновалентных форматах IgG с CD28 человека на клетках. Медианную интенсивность флуоресценции для связывания с клетками СНО, экспрессирующими CD28 человека (родительская линия клеток CHO-k1 АТСС № CCL-1, модифицированная для стабильной сверхэкспрессии CD28 человека), в сравнении с отрицательным контролем (анти-DP47) и оригинальным TGN1412 оценивали методом проточной цитометрии. Кривые связывания вариантов 1-10 показаны на Фиг. 4А, кривые вариантов 11-22 - на Фиг. 4Б кривые вариантов 23-31 - на Фиг. 4В. Изображены два технических повтора с СО.
На Фиг. 4Г и 4Д показано связывание нацеленных на FAP вариантов биспецифического агонистического антитела к CD28 в формате hulgG1 PG-LALA 1+1 с выбранными вариантами агонистического антитела к CD28 со сниженной аффинностью с CD28 на клетках. Кривые связывания биспецифических конструкций 1+1 с вариантами 8, 11, 12, 15, 16 и 17 показаны на Фиг. 4Г, тогда как кривые связывания биспецифических конструкций 1+1 с вариантами 19, 23, 25, 27 т 29 показаны на Фиг. 4Д. Выбранные связывающие молекулы были отобраны на основании аффинности для получения в биспецифической нацеленном на FAP формате 1+1. На Фиг. 4Е и 4Ж показано связывание одних и тех же нацеленных на FAP вариантов биспецифического агонистического антитела к CD28 в формате huIgG1 PG-LALA 1+1 с FAP человека. Приведена медианная интенсивность флуоресценции для связывания с клетками СНО, экспрессирующими CD28 человека (родительская линия клеток CHO-k1 АТСС № CCL-61, модифицированная для стабильной сверхэкспрессии CD28 человека), или клетками 3Т3, экспрессирующими FAP человека (линия клеток NIH/3T3 (АТСС CRL-1658)), в сравнении с отрицательным контролем (анти-DP47) и TGN1412 (молекула А), которую оценивали методом проточной цитометрии. Приведены три технических повтора с СПС.
In vitro активность выбранных нацеленных на FAP вариантов биспецифического агонистического антитела к CD28 в формате huIgG1 PG-LALA 1+1 проиллюстрирована на Фиг. 4И, 4К и 4Л. Т-клетки МКПК инкубировали с MCSP- и FAP-экспрессирующими клетками меланомы MV3 в течение 5 суток в присутствии ограничивающей концентрации MCSP-TCB (5 пМ, P1AD2189) и возрастающих концентраций конструкций FAP-CD28 с указанными вариантами CD28-связывающих молекул. На Фиг. 4И показано разведение CFSE как показатель пролиферации CD8 Т-клеток, которую оценивали методом проточной цитометрии. Планки погрешностей представляют СПС, на графиках приведены три технических повтора репрезентативных результатов от 2 доноров. На Фиг. 4К показана корреляция KD (нМ) варианта CD28-связывающей молекулы в отношении активности по площади под кривой (а) как % от родительского клона TGN1412 (CD28 (SA)). На Фиг. 4Л показано уничтожение клеток-мишеней через 90 ч.
Фиг. 5А-5Г относятся к созданию предварительной культуры CD28(SA) с высокой плотностью (ВП). Т-клетки МКПК предварительно культивировали при высокой плотности (ВП) в течение 2 суток или использовали свежими после выделения МКПК и стимулировали возрастающими концентрациями CD28(SA). Изображено разведение CFSE как показатель пролиферации Т-клеток через 5 суток после стимуляции CD28(SA) (молекула А, Р1АЕ1975) (Фиг. 5А) и секреции цитокинов после 2 суток (Фиг. 5Б) стимуляции. На Фиг. 5В показано процентное значение экспрессии FcγRIIb в моноцитах МКПК и В-клетках до и после 2 суток предварительного культивирования МКПК ВП, которое оценивали методом проточной цитометрии. Фиг. 5Г: МКПК ВП после предварительного культивирования культивировали совместно с CD28(SA) в течение 5 суток в присутствии или отсутствие блокирующего антитела FcγRIIb или изотипического контроля и оценивали процентное значение разведения CFSE CD4 Т-клеток методом проточной цитометрии. Графики представляют по меньшей мере 6 доноров (Фиг. 5А, 5Б) и 2 доноров (Фиг. 5В, 5Г), каждого из которых оценивали в независимых экспериментах. На графиках показаны три технических повтора. Планки погрешностей указывают СПС. Статистический анализ проводили с помощью t-критерия Стьюдента. ***: p<0,001. Суперагонизм CD28(SA) IgG4 зависит от перекрестного связывания с FcγRIIb.
На Фиг. 6А и 6Б показана пролиферация Т-клеток, т.е. разведение CFSE CD4 Т-клеток через 5 суток после стимуляции оригинальным Fc IgG4 дикого типа CD28(SA) (Р1АЕ1975) или CD28(SA), несущим мутацию P329G-LALA (P1AD9289). Т-клетки предварительно культивировали при высокой плотности в течение 2 суток. Графики представляют по меньшей мере 3 независимых эксперимента. Приведены три технических повтора. Fc-сайленсинг устраняет суперагонизм в TGN1412. Добавление нацеленного на опухоль фрагмента к TGN1412 с Fc-сайленсингом восстанавливает суперагонизм, который затем зависит от наличия опухолевой мишени.
На Фиг. 7А, 7Б, 7В и 7Г показано сравнение нацеленных на FAP агонистов CD28 в разных форматах (2+2 и 1+2) и с суперагонистическими (CD28 (SA)) связывающими молекулами и стандартными агонистическими связывающими молекулами (9.3, CD28 (CA)). Нацеленные на FAP агонисты CD28 со стандартными агонистическими CD28-связывающими молекулами не функционируют как суперагонисты. Т-клетки МКПК культивировали совместно с клетками 3Т3-huFAP (присутствует FAP) в присутствии возрастающих концентраций форматов FAP-CD28 с суперагонистическими связывающими молекулами (SA, Фиг. 7А) или стандартными агонистическими связывающими молекулами (9.3, Фиг. 7Б) в течение 5 суток, показана пролиферация Т-клеток. Потом Т-клетки МКПК также культивировали совместно с клетками 3Т3 ДТ (отсутствует FAP) в присутствии возрастающих концентраций форматов FAP-CD28 с суперагонистическими связывающими молекулами (SA, Фиг. 7В) или стандартными агонистическими связывающими молекулами (9.3, Фиг. 7Г) в течение 5 суток. Изображено разведение CFSE как показатель пролиферации CD8 Т-клеток, оцениваемой методом проточной цитометрии на 5 сутки после стимуляции. На графиках показаны кумулятивные данные по 3 донорам в 3 независимых экспериментах. Планки погрешностей показывают СПС. В таких же экспериментальных условиях измеряли количество цитокинов в супернатантах после 2 суток совместного культивирования. Значения приведены на Фиг. 7Д.
Способность FAP-CD28 в различных форматах с суперагонистическими связывающими молекулами CD28(SA) или стандартными агонистическими связывающими молекулами (CD28(CA)) индуцировать уничтожение FAP-экспрессирующих клеток меланомы RFP-MV3 оценивали в течение 90 ч с помощью визуализации живых клеток, используя технологию IncuCyte. Все молекулы, включая FAP-TCB (P1AD4645), использовали в концентрации 10 нМ. На Фиг. 8А, 8Б и 8В показаны репрезентативные результаты от трех доноров с тремя техническими повторами, соответственно. На Фиг. 8Г показаны кумулятивные результаты, выраженные как площадь под кривой (ППК) в момент t=90 ч, по 3 донорам в 3 независимых экспериментах. Прямоугольниками показаны 25-ый - 75-ый процентили, усами показаны значения от мин до макс. Статистический анализ проводили с помощью спаренного 1-факторного ANOVA. ***: p<0,001, нз: не значимо.
Сравнение нацеленных на СЕА агонистов CD28 в разных форматах с суперагонистическими и стандартными агонистическими связывающими молекулами показано на Фиг. 9А и 9Б. Способность CEA-CD28 в различных форматах с суперагонистическими связывающими молекулами CD28(SA) или стандартными агонистическими связывающими молекулами (CD28(CA)) индуцировать уничтожение СЕА-экспрессирующих клеток рака желудка RFP+MKN45 оценивали в течение 90 ч с помощью визуализации живых клеток, используя технологию IncuCyte. Все молекулы, включая СЕАСАМ5-ТСВ (P1AD5299), использовали в концентрации 10 нМ. На Фиг. 9А показаны репрезентативные результаты от одного донора с тремя техническими повторами. На Фиг. 9Б показан статистический анализ трех технических повторов, выраженный как площадь под кривой (ППК) в момент t=90 ч, по 1 донору в 1 эксперименте. Прямоугольниками показаны 25-ый - 75-ый процентили, усами показаны значения от мин до макс.Статистический анализ проводили с помощью спаренного 1-факторного ANOVA. ***: р<0,001. Показано, что нацеленные на СЕА агонисты CD28 со стандартными агонистическими CD28-связывающими молекулами не проявляют суперагонистического поведения.
На Фиг. 10А, 10Б и 10В показано, что нацеленные агонисты CD28 с одновалентными суперагонистическими связывающими молекулами не функционируют суперагонистически. Т-клетки МКПК совместно культивировали в течение 5 суток с клетками 3Т3-huFAP в присутствии возрастающих концентраций FAP-CD28 с двухвалентными CD28-связывающими молекулами (P1AD9011, закрашенные круги) или FAP-CD28 с одновалентностью в отношении связывания CD28 (P1AD4492, незакрашенные круги). На Фиг. 10А показано разведение CFSE CD8 Т-клеток. Кроме того, активацию Т-клеток оценивали путем обнаружения маркеров активации CD69 (Фиг. 10Б) и CD25 (Фиг. 10В) методом проточной цитометрии. Показана средняя интенсивность флуоресценции (СИФ) окрашивания CD69 и CD25 через 5 суток после стимуляции. Показаны три технических повтора по 1 донору, планки погрешностей указывают СПС. Показано, что для TGN1412-подобного суперагонизма необходимо одновалентное связывание CD28.
На Фиг. 11А и 11Б показано, что в комбинации с Т-клеточными биспецифическими антителами (ТСВ) ТСВ-опосредованные эффекторные функции поддерживаются одновалентным и двухвалентными связыванием CD28 нацеленных на FAP агонистических CD28-антигенсвязывающих молекул со сравнимой активностью, но необходима одновалентность CD28-связывающей молекулы для сохранения зависимости от опухолевой мишени для агонистов CD28 в присутствии ТСВ. На Фиг. 11А в случае наличия FAP Т-клетки МКПК инкубировали с MCSP- и FAP-экспрессирующими клетками меланомы MV3 в течение 90 ч в присутствии комбинированной ограничивающей концентрации MCSP-TCB (5 пМ, P1AD2189) и возрастающей концентрации (диапазон 0-10 нМ) FAP-CD28(SA) с двухвалентным или одновалентным связыванием с CD28, соответственно. Изображено уничтожение клеток-мишеней через 90 ч посредством визуализации живых клеток с использованием технологии IncuCyte. На Фиг. 11Б, Т-клетки МКПК в течение 90 ч совместно культивировали с FAP-отрицательными СЕА-экспрессирующими клетками рака желудка MKN45 (отсутствует FAP) в присутствии ограничивающих концентраций СЕАСАМ5-ТСВ (10 пМ, P1AD5299) в комбинации с возрастающей концентрацией (диапазон 0-10 нМ) FAP-CD28 с двухвалентным или одновалентным связыванием с CD28, соответственно. Изображено уничтожение клеток-мишеней через 90 ч по оценке IncuCyte. Данные показывают уничтожение клеток-мишеней MKN45 в течение времени от 1 донора в 1 эксперименте, три технических повтора, планки погрешностей указывают СПС.
На Фиг. 12А и 12Б показано, что FAP-CD28(SA) с двухвалентным связыванием с CD28 утрачивает FAP-зависимость при комбинации с биспецифическими Т-клеточными антителами. На Фиг. 12А ТСВ отсутствует. Т-клетки МКПК культивировали совместно с СЕА-экспрессирующими MKN45 и 3Т3-huFAP (условие «присутствует FAP», закрашенные круги) или 3T3-WT (условие «отсутствует FAP», незакрашенные круги), соответственно, в присутствии возрастающих концентраций FAP-CD28(SA) 2+1. Комбинация с ТСВ показана на Фиг. 12Б. Т-клетки МКПК культивировали совместно с СЕА-экспрессирующими MKN45 и 3Т3-huFAP (условие «присутствует FAP», закрашенные круги) или 3T3-WT (условие «отсутствует FAP», незакрашенные круги), соответственно, в присутствии ограничивающих концентраций СЕАСАМ5-ТСВ (10 пМ, P1AD5299) и возрастающих концентраций FAP-CD28 2+1 SA. Показана пролиферация CD8 Т-клеток через 5 суток после стимуляции. Данные представляют 2 независимых эксперимента с 2 донорами. Показаны результаты по одному донору, данные представляют три технических повтора, планки погрешностей указывают СПС.
На Фиг. 13А, 13Б и 13 В показана функциональность антигенсвязывающих молекул FAP-CD28(SA) с одновалентным связыванием с CD28 в разных форматах. Молекула С представляет собой FAP-CD28(SA) в классическом формате 1+1 (P1AD4492), молекула Н представляет собой FAP-CD28(SA) в формате 1+1 «голова к хвосту» (Н2Т) (Р1АЕ2021), молекула I представляет собой FAP-CD28(SA) в формате 1+1 с С-концевым слиянием FAP-связывающей молекулы (Р1АЕ2236), а молекула G представляет собой FAP-CD28(SA) в формате 2+1 (P1AD5231). В качестве референса использовали двухвалентную С028-антигенсвязывающую молекулу (P1AD9011). Т-клетки МКПК инкубировали с MCSP- и FAP-экспрессирующими клетками меланомы МУЗ в присутствии ограничивающей концентрации MCSP-TCB (5 пМ, P1AD2189) и возрастающих концентраций (диапазон 0 10 нМ) FAP-CD28 в заданных форматах. Изображено разведение CFSE как показатель пролиферации Т-клеток для CD8 (Фиг. 13А) и CD4 Т-клеток (Фиг. 13Б) через 5 суток, оцениваемой методом проточной цитометрии. Фиг. 13 В: Показано уничтожение клеток MV3 в течение 84 часов в присутствии только 5 пМ MCSP-TCB по сравнению с комбинацией 5 пМ MCSP-TCB и возрастающих концентраций FAP-CD28 в различных форматах. Уничтожение оценивали посредством визуализации живых клеток, используя систему IncuCyte. Все молекулы были способны поддерживать ТСВ-опосредованные эффекторные функции. На графиках приведены кумулятивные данные по 3 независимым экспериментам и 4 донорам. 10 пМ MCSP-TCB; Э:М 20; статистика: 2-факторный ANOVA. Звездочками указана наименьшая концентрация, при которой дополнение является значимым по сравнению с одним ТСВ: *р≤0,05, **р≤0,01; ***p≤0,001. Планки погрешностей указывают СПС.
Уничтожение клеток-мишеней CEA-CD28 в формате 1+1 в комбинации с ТСВ показано на Фиг. 14. Т-клетки МКПК в течение 90 ч культивировали совместно с СЕА-экспрессирующими клетками рака желудка MKN45 в присутствии ограничивающих концентраций СЕАСАМ5-ТСВ (10 пМ, P1AD5299) в комбинации с 2 нМ CEA-CD28 (P1AE3J27) или ненацеленным CD28 (P1AD8944). Данные показывают уничтожение клеток-мишеней MKN45 в течение времени от 1 донора в 1 эксперименте. Уничтожение оценивали посредством визуализации живых клеток, используя систему IncuCyte. Показано, что только комбинация приводит к уничтожению клеток-мишеней, при заданной концентрации молекулы отдельно не индуцируют уничтожение. CEA-CD28 демонстрирует синергизм с СЕАСАМ5-ТСВ.
На Фиг. 15 показано, что CEA-CD28 усиливает СЕА-ТСВ и СЕАСАМ5-ТСВ и снижает порог экспрессии СЕА для ТСВ для индукции активации Т-клеток. Т-клетки МКПК инкубировали с возрастающими концентрациями СЕА-ТСВ (P1AD4646) или СЕАСАМ5-ТСВ (P1AD5299) и фиксированными концентрациями CEA-CD28 (Р 1АЕ3127) в присутствии линий клеток-мишеней с разными уровнями экспрессии СЕА: (i) MKN45 (высокая экспрессия, прибл. 400000 СЕА-связывающих сайтов/клетка), (ii) Lovo (средняя экспрессия, прибл. 60000 СЕА-связывающих сайтов/клетка), (in) HT-29 (низкая экспрессия, прибл. 6000 СЕА-связывающих сайтов/клетка). Пролиферацию Т-клеток оценивали как показатель активации Т-клеток методом проточной цитометрии.
Связывание выбранных вариантов С028-связывающих молекул со сниженной аффинностью в биспецифическом СЕА-нацеленном одновалентном формате 1+1 с CD28 на клетках показано на Фиг. 16. Медианную интенсивность флуоресценции для связывания антител CEA-CD28 или анти-DP47 антитела (отрицательный контроль) с клетками СНО, экспрессирующими CD28 человека (родительская линия клеток CHO-k1 ATCC №CCL-61, модифицированная для стабильной сверхэкспрессии CD28 человека), оценивали методом проточной цитометрии. Приведены три технических повтора с СПС.
На Фиг. 17А, 17Б и 17В показана функциональность выбранных вариантов CD28-связывающих молекул со сниженной аффинностью в биспецифическом СЕА-нацеленном одновалентном формате 1+1. Т-клетки МКПК культивировали совместно с СЕА-экспрессирующими клетками рака желудка MKN45 в присутствии ограничивающих концентраций СЕАСАМ5-ТСВ (10 пМ, P1AD5299) в комбинации с 2 нМ молекул CEA-CD28 1+1 с вариантами CD28-связывающих молекул. Пролиферацию CD8 Т-клеток (Фиг. 17А) и пролиферацию CD4 Т-клеток (Фиг. 17Б) оценивали путем разведения CFSE методом проточной цитометрии после 5 суток совместного культивирования. Уничтожение клеток-мишеней оценивали после 90 ч инкубации (Фиг. 17С). Данные показывают уничтожение клеток-мишеней MKN45 в течение времени от 1 донора в 1 эксперименте. Все молекулы способны поддерживать СЕАСАМ5-ТСВ-опосредованные эффекторные функции.
На Фиг. 18 показано связывание гуманизированных вариантов СЕА(А5 В7) hulgGI P329G LALA с MKN-45 по сравнению со связыванием родительского мышиного антитела А5 В7. Обнаружение антител проводили с помощью флуоресцентно меченного вторичного антитела, а флуоресценцию измеряли методом проточной цитометрии.
На Фиг. 19А-19В приведены схематические иллюстрации рекомбинантных белков, демонстрирующие разные домены белка СЕАСАМ5, которые использовали в качестве антигенов в методе фагового дисплея. На Фиг. 19А показана конструкция NABA-avi-His, состоящая ищ 4 Ig-подобных доменов N, А1, В и А2. На Фиг. 19Б показана конструкция N(A2B2)A-avi-His, а на Фиг. 19 В проиллюстрирована конструкция NA(B2)A-avi-His.
На Фиг. 20А и 20Б показаны последовательности VH и VL, соответственно, гуманизированного антитела к СЕА A5H1EL1D, в которой рандомизированные позиции обозначены X.
Схематические изображения фаговых векторов библиотек созревания аффинности показаны на Фиг. 21А (библиотека созревания аффинности CDRH1/H2), Фиг. 21Б (библиотека созревания аффинности CDRL1/H2) и Фиг. 21 В (библиотека созревания аффинности CDRH3/CDRL3).
На Фиг. 22А и 22Б показано выравнивание аминокислотных последовательностей VH (Фиг. 22А) и аминокислотных последовательностей VL (Фиг. 22Б) гуманизированных вариантов антитела CEA(A5H1EL1D) с созревшей аффинностью.
На Фиг. 23А-23Г показаны схематические иллюстрации биспецифических СЕА/CD28-антигенсвязывающих молекул, описанных в примере 11.
На Фиг. 23А показана биспецифическая СЕА-CD28-антигенсвязывающая молекула в формате 1+1, в которой СЕА-антигенсвязывающий домен представлен как кросс-Fab (обмен VH/VL) и в Fab-фрагменте, несущем CD28-антигенсвязывающий домен, присутствуют заряженные модификации с целью поддержания правильного спаривания легких цепей. Fc-домен имеет модификации типа «выступ во впадину» и мутации P329G LALA для устранения связывания с Fcγ-рецепторами. На Фиг. 23Б CD28-антигенсвязывающий домен представлен как кросс-Fab (обмен VH/VL), a Fab-фрагмент, несущий СЕА-антигенсвязывающий домен, содержит заряженные модификации.
На Фиг. 23В проиллюстрирована биспецифическая CEA-CD28-антигенсвязывающая молекула в формате 2+1, в которой CD28-антигенсвязывающий домен представлен как кросс-Fab, а два Fab-фрагмента с СЕА-антигенсвязывающими доменами слиты друг с другом посредством тяжелой цепи (голова к хвосту).
На Фиг. 23Г проиллюстрирована биспецифическая антигенсвязывающая молекула CEA-CD28 в формате 2+1, в которой CD28-антигенсвязывающий домен представлен кросс-Fab, который слит в своем С-конце с N-концом другой тяжелой цепи «двухвалентного» антитела к СЕА («классический» формат).
На Фиг. 24 показано, что анти-СЕА клон Р002.139 с созревшей аффинностью демонстрирует улучшенное связывание с СЕАСАМ5 на СЕА-экспрессирующих клетках MV3. Показано связывание биспецифических антител CEA-CD28, несущих анти-СЕА клон Р002.139 с созревшей аффинностью или родительский клон A5H1EL1D. Медианную интенсивность флуоресценции для связывания биспецифических антител CEA-CD28 или анти-DP47 антитела (отрицательный контроль) с клетками MV3, генетически сконструированными для экспрессии человеческого СЕАСАМ5, оценивали методом проточной цитометрии. Приведены два технических повтора с СПС. График представляет 3 независимых эксперимента.
На Фиг. 25А и 25Б показано, что анти-СЕА клон Р002.139 с созревшей аффинностью демонстрирует улучшенную функциональность в репортерном анализе с IL-2. Показаны результаты регистрации люминесценции после 6 ч совместной инкубации клеток MKN45, репортерных клеток IL-2 с 5 нМ СЕА-ТСВ и CEA-CD28, несущим клон Р002.139 с созревшей аффинностью или родительский клон A5H1EL1D. На Фиг. 25А показана зависимость доза - ответ. Пунктирной линией указана люминесценция, обеспечиваемая одним СЕА-ТСВ. На Фиг. 25Б показаны значения площади под кривой, рассчитанные по данным, приведенным на Фиг. 25А. Приведены два технических повтора с СПС. График представляет 3 независимых эксперимента.
На Фиг. 26 показан дизайн исследования эффективности с биспецифическими антителами CEA-CD28 (сравнение разных клонов СЕА) в комбинации с СЕА ТСВ у гуманизированных мышей с ксенотрансплантатом MKN45. Показаны дизайн и разные группы лечения.
На Фиг. 27А-27Д показаны результаты исследования эффективности с комбинацией CEA-CD28 и СЕА ТСВ у гуманизированных мышей с ксенотрансплантатом MKN45. Показаны средний объем опухолей (Фиг. 27А) или рост опухолей у отдельных мышей для четырех групп обработки, отложенные по оси у (Фиг. 27Б-27Д). На Фиг. 27Б показан рост опухоли для каждой отдельной мыши в группе носителя, на Фиг. 27В - для мышей, обработанных одним СЕА ТСВ, на Фиг. 27Г - для мышей, обработанных СЕА ТСВ и CEA(T84.66)-CD28 (SA_вариант 15) и на Фиг. 27Д - для мышей, обработанных СЕА ТСВ и CEA(A5H1EL1D)-CD28 (SA вариант 15). Можно видеть повышение ТСВ-опосредованной регрессии опухолей в присутствии обоих биспецифических антител к CEA-CD28.
На Фиг. 28 показан дизайн исследования эффективности с биспецифическими антителами CEA-CD28 (сравнение разных клонов CD28) в комбинации с СЕАСАМ5 ТСВ у гуманизированных мышей с ксенотрансплантатом ВХРС3. Показаны дизайн и разные группы лечения.
На Фиг. 29 показана кинетика роста опухолей (среднее, +СПС) для всех групп обработки, соответствующие значения ИРО для каждой группы обработки приведены в таблице 33 (пример 13.2).
Ex vivo иммуно-ФД данные приведены на Фиг. 30А-30Г. На Фиг. 30А приведены репрезентативные точечные графики (CD3 против CD45 и CD4 против CD8) окрашенных суспензий одиночных опухолевых клеток для каждой группы обработки. Обобщенные данные по инфильтрации CD3, CD8 и CD4 Т-клеток приведены на Фиг. 30Б (CD3), Фиг. 30 В (CD8) и Фиг. 30Г (CD4), соответственно.
На Фиг. 31 показан дизайн исследования эффективности с биспецифическим антителом CEA-CD28 (CEA(A5H1EL1D)-CD28 (SA_вариант 8)) в комбинации с СЕА ТСВ у гуманизированных мышей с ксенотрансплантатом MKN45. Показаны дизайн и разные группы лечения.
На Фиг. 32 показана кинетика роста опухолей (среднее, +СПС) для всех групп обработки, соответствующие значения ИРО для каждой группы обработки приведены в таблице 35 (пример 13.3).
Ex vivo иммуно-ФД данные приведены на Фиг. 33А и 33Б. На Фиг. 33А приведены репрезентативные точечные графики окрашенных суспензий одиночных опухолевых клеток для каждой группы обработки. Обобщенные данные по инфильтрации CD3+ Т-клеток приведены на Фиг. 33Б.
На Фиг. 34А-34Г показаны схематические иллюстрации биспецифических CD28-антигенсвязывающих молекул, описанных в примере 14.
На Фиг. 34А показана биспецифическая EpCAM-CD28-антигенсвязывающая молекула в формате 1+1, в которой CD28-антигенсвязывающий домен представлен как кросс-Fab (обмен VH/VL) и в Fab-фрагменте, несущем ЕрСАМ-антигенсвязывающий домен, присутствуют заряженные модификации с целью поддержания правильного спаривания легких цепей. Fc-домен имеет модификации типа «выступ во впадину» и мутации P329G LALA для устранения связывания с Fcγ-рецепторами.
На Фиг. 34Б Fab, несущий CD28-антигенсвязывающий домен, содержит заряженные модификации, a Fab, несущий HER3-антигенсвязывающий домен, представлен как кросс-Fab (обмен VH/VL).
На Фиг. 34В проиллюстрирована биспецифическая CD30-CD28-антигенсвязывающая молекула в формате 1+1, в которой молекула Fab, несущая CD28-антигенсвязывающий домен, содержит заряженные модификации, a Fab, несущий CD30-антигенсвязывающий домен, представлен как кросс-Fab (обмен VH/VL).
На Фиг. 34Г проиллюстрирована биспецифическая TPBG-CD28-антигенсвязывающая молекула в формате 1+1, в которой молекула Fab, несущая CD28-антигенсвязывающий домен, содержит заряженные модификации, a Fab, несущий TPBG (5Т4)-антигенсвязывающий домен, представлен как кросс-Fab (обмен VH/VL).
Фиг. 35А-35В относятся к функциональным характеристикам биспецифических антигенсвязывающих молекул EpCAM-CD28. На Фиг. 35А показано, что EpCAM-CD28 (молекула 14А) связывается с CD28 человека на клетках CHO-k1, экспрессирующих CD28, по оценке методом проточной цитометрии. Связывание ЕрСАМ на клетках НТ29 по оценке методом проточной цитометрии показано на Фиг. 35Б. Анти-DP47 служил в качестве отрицательного контроля для неспецифического связывания соединений антител с клетками. Точки представляют средние значения по двух техническим повторам. На Фиг. 35 В показано, что EpCAM-CD28 (Р1АЕ9051) усиливает Т-клеточные ответы на анти-CD3 стимул в репортерном анализе с IL-2. Показана активация репортерных клеток IL-2, измеренная по результата люминесценции после 6 часов совместной инкубации с НТ-29 в присутствии субоптимальных концентраций анти-CD3 IgG (10 нМ) и возрастающих концентраций EpCAM-CD28. Точки представляют средние значения по двух техническим повторам.
Фиг. 36А-36В относятся к функциональным характеристикам биспецифических антигенсвязывающих молекул HER3-CD28. На Фиг. 36А показано, что HER3-CD28 (P1AF0151) связывается с CD28 человека на клетках CHO-k1, экспрессирующих CD28, по оценке методом проточной цитометрии. Связывание HER3-CD28 с HER3 на клетках T-47D по оценке методом проточной цитометрии показано на Фиг. 36Б. Анти-DP47 служил в качестве отрицательного контроля для неспецифического связывания соединений антител с клетками. Точки представляют средние значения по двух техническим повторам. На Фиг. 36 В показано, что HER3-CD28 (P1AF0151) усиливает Т-клеточные ответы на анти-CD3 стимул в репортерном анализе с IL-2. Показана активация репортерных клеток IL-2, измеренная по результата люминесценции после 6 часов совместной инкубации с клетками T-47D в присутствии субоптимальных концентраций анти-CD3 IgG клона OKT3 (10 нМ) и возрастающих концентраций HER3-CD28. Точки представляют средние значения по двух техническим повторам.
На Фиг. 37А-37В показаны схематические иллюстрации биспецифических CD28-антигенсвязывающих молекул, нацеленных на антиген клеточной поверхности множественной миеломы (ММ), описанных в примере 16.
На Фиг. 37А показана биспецифическая GPRC5D-CD28-антигенсвязывающая молекула в формате 1+1, в которой CD28-антигенсвязывающий домен представлен как кросс-Fab (обмен VH/VL) и в Fab-фрагменте, несущем GPRC5D-антигенсвязывающий домен, присутствуют заряженные модификации с целью поддержания правильного спаривания легких цепей. Fc-домен имеет модификации типа «выступ во впадину» и мутации P329G LALA для устранения связывания с Fcγ-рецепторами.
На Фиг. 37Б Fab, несущий CD28-антигенсвязывающий домен, содержит заряженные модификации, a Fab, несущий CD38-антигенсвязывающий домен, представлен как кросс-Fab (обмен VH/VL).
На Фиг. 37В проиллюстрирована биспецифическая BCMA-CD28-антигенсвязывающая молекула в формате 1+1, в которой молекула Fab, несущая CD28-антигенсвязывающий домен, представлена как кросс-Fab (обмен VH/VL), a Fab, несущий ВСМА-антигенсвязывающий домен, содержит заряженные модификации.
На Фиг. 37Г проиллюстрировано анти-GPRC5D/анти-CD3 биспецифическое антитело (GPRC5D ТСВ) в формате 2+1, в которой молекулы Fab, несущие GPCR5D-антигенсвязывающий домен, содержат заряженные модификации, a Fab, несущий CD3-антигенсвязывающий домен, представлен как кросс-Fab (обмен VH/VL).
Фиг. 38А-38Е относятся к связыванию биспецифических антигенсвязывающих молекул, нацеленных на CD28 и антиген клеточной поверхности множественной миеломы (ММ), с клетками (пример 17.1). Показано связывание биспецифических антигенсвязывающих молекул с CD28 человека на клетках СНО huCD28 с145 (Фиг. 38А и 38Д), с CD38 человека на клетках OCI-Ly18 (Фиг. 38Б), ВСМА человека (антиген созревания В-клеток, Фиг. 38В) на клетках IM-9 и с GPRC5D человека на клетках СНО huGPRC5D L2 (Фиг. 38Г и 38Е), экспрессируемым в указанных линиях клеток. Приведены относительные медианные значения флуоресценции (МИФ) с двух повторов с СО. Значения ЕС50 для связывания были рассчитаны GraphPadPrism и включены в таблицу 38.
Активация Т-клеток для биспецифических антигенсвязывающих молекул, нацеленных на CD28 и антиген клеточной поверхности множественной миеломы (ММ), оцениваемая в репортерном анализе с IL-2, показана на Фиг. 39А-39Е. Показан репортерный анализ с IL-2 после 5 и 22 часов инкубации с определением по люминесценции. IL2-репортерные эффекторные клетки и GPRC5D-экспрессирующие клетки-мишени инкубировали в соотношении эффектора к мишени (Э:М) 5:1. GPRC5D-TCB добавляли в фиксированной конечной аналитической концентрации 1 нМ, указанные нацеленные на ММ биспецифические CD28-антигенсвязывающие молекулы титровали, как указано. Репрезентативные кривые доза ответ приведены для CD38-CD28 на Фиг. 39А (после 5 часов инкубации) и Фиг. 39Б (после 22 часов), для BCMA-CD28 на Фиг. 39В (после 5 часов) и Фиг. 39Г (после 22 часов) и для GPRC5D-CD28 на Фиг. 39Д (после 5 часов) и Фиг. 39Е (после 22 часов инкубации).
На Фиг. 40А-40В показано усиление опосредованного Т-клетками лизиса GPRC5D-экспрессирующей линии клеток MM NCI-H929 в присутствии 0,2 нМ указанных биспецифических молекул к CD28 CD38-CD28 (Фиг. 40А), BCMA-CD28 (Фиг. 40Б) и GPRC5D-CD28 (Фиг. 40В). Лизис определяли после совместной инкубации человеческих пан-Т-клеток и опухолевых клеток-мишеней ММ в конечном соотношении Э:М 1:1 в течение 22 часов. Изображены два технических повтора с СО. Значения ЕС50 и значения площади под кривой для лизиса опухолевых клеток были рассчитаны GraphPadPrism и приведены в таблице 39.
На Фиг. 41А показана схематическая иллюстрация биспецифической CD19-CD28 антигенсвязывающей молекулы в формате 1+1, описанной в примере 18, в которой в Fab, содержащем СD19-антигенсвязывающий домен, домены VH и VL заменены друг другом (VH/VL кросс-fab) и в которой в Fab, содержащем CD28-антигенсвязывающий домен, определенные аминокислоты в домене СН1 и CL обменены (зарядовые варианты) для обеспечения лучшего спаривания с легкой цепью. На Фиг. 41В показана соответствующая молекула, в которой CD19-антигенсвязывающий домен был замещен CD79b-антигенсвязывающим доменом (анти-CB79b кросс-fab).
Фиг. 42 относится к определению кинетических и термодинамических параметров CD79b (полатузумаб) в конструкции CD79b (huMA79b.v28) - CD28 (v15) 1+1. Проводили захват растворимого рекомбинантного CD79b-His на чипе СМ5 с помощью анти-пента-His антитела, а биспецифическое антитело к CD79b (huMA79b.v28) - CD28 (v15) 1+1 использовали как аналит. Сглаженные линии представляют глобальную аппроксимацию данных 1:1 моделью взаимодействия.
На Фиг. 43А приведена медианная интенсивность флуоресценции (МИФ) для связывания варианта 15 CD19-CD28 (Р1АЕ9040) с четырьмя разными линиями В-клеток, экспрессирующими разные уровни CD19. Связывание оценивали методом проточной цитометрии. Приведены два технических повтора с СПС. На Фиг. 43Б изображено окрашивание FACS в отношении CD19 четырех разных линий В-клеток (МИФ).
Связывание CD19-CD28 с различной аффинностью CD28 к CD19 и CD28 человека на клетках показано на Фиг. 44А и 44Б. Приведена медианная интенсивность флуоресценции (МИФ) для связывания с клетками CHOk1-CD28 (Фиг. 44А) и для связывания CD19 на В-клетках Nalm6 (Фиг. 44Б). Точки представляют два технических повтора с СПС. Соответствующие значения ЕС5о приведены в таблице 42 (CHOk1-CD28) и таблице 43 (Nalm6) примера 20. Связывание оценивали методом проточной цитометрии.
На Фиг. 45А-45Г показано, что CD19-CD28v15 усиливает CD20-TCB в репортерном анализе с IL-2 в присутствии разных линий В-клеток. Показана активация репортерных клеток IL-2, измеренная по результата люминесценции (ЛЮМ) после 6 часов совместной инкубации с разными линиями В-клеток в присутствии субоптимальных концентраций CD20-TCB и возрастающих концентраций CD19-CD28v15. Точки представляют два технических повтора с СПС.Субоптимальная концентрация CD20-TCB отличается для линий клеток-мишеней: 10 нМ для Nalm6, 0,05 нМ для RCK8, WSU DLCL2 и Z138.
На Фиг. 46 проиллюстрировано, что CD19-CD28 с различной аффинностью CD28 усиливает опосредованную CD20-TCB активацию Т-клеток. Показана активация репортерных клеток IL-2, измеренная по результата люминесценции (ЛЮМ) после 6 часов совместной инкубации с В-клетками Nalm6 в присутствии субоптимальных концентраций CD20-TCB (10 нМ) и возрастающих концентраций CD19-CD28v15. Точки представляют два технических повтора с СПС.
Оценивали статус активации полученных из МКПК Т-клеток после совместного культивирования с CD20-экспрессирующими клетками-мишенями (Nalm6) (соотношение Э:М 5:1) и CD19-CD28 в отсутствие или присутствии CD20-TCB. Активность CD19-CD28 в отсутствие или присутствии сигналов ТКР показана на Фиг. 47. Показана экспрессия CD69 полученных из МКПК CD4 Т-клеток после 48 ч совместной инкубации с клетками Nalm6, возрастающими концентрациями CD19-CD28v15 в присутствии или отсутствие 10 нМ CD20-TCB. Данные представляют три технических повтора с СПС.
На Фиг. 48А-48Г проиллюстрировано, что один CD19-CD28 не индуцирует секрецию цитокинов в МКПК. Показано высвобождение цитокинов в цельных МКПК после 48 часов совместного культивирования с молекулами CD19-CD28 в присутствии или отсутствие CD20-TCB. Планки представляют среднее +СПС по трем техническим повторам. Представлены данные по 2 донорам. Секрецию цитокинов оценивали с помощью 17-канального анализа цитокинов человека Bio-Plex Pro. Показаны IFNγ (Фиг. 48А), IL-2 (Фиг. 48Б), IL-10 (Фиг. 48В) и TNF (Фиг. 48Г).
На Фиг. 49А и 49Б приведены функциональные данные, относящиеся к CD79b-CD28, который усиливает CD20-TCB в репортерном анализе с IL-2 в присутствии В-клеток Z138. На Фиг. 49А приведена медианная интенсивность флуоресценции (МИФ) для связывания с CD79b на В-клетках Z138. На Фиг. 49Б показано, что CD79b-CD28 усиливает CD20-TCB в репортерном анализе с IL-2 в присутствии В-клеток Z138. Показана активация репортерных клеток IL-2, измеренная по результата люминесценции (ЛЮМ) после 6 часов совместной инкубации с разными линиями В-клеток в присутствии субоптимальных концентраций CD20-TCB и возрастающих концентраций CD79b-CD28. Точки представляют два технических повтора с СПС.
На Фиг. 50 показан дизайн исследования эффективности с биспецифическими антителами CD19-CD28 (сравнение двух разных клонов CD28) у гуманизированных мышей с ксенотрансплантатом NALM6. Показаны дизайн и разные группы лечения.
На Фиг. 51А-51Г показаны результаты исследования эффективности с CD19-CD28 у гуманизированных мышей с ксенотрансплантатом NALM6. Показаны средний объем опухолей (Фиг. 51А) или рост опухолей у отдельных мышей для трех групп обработки, отложенные по оси у (Фиг. 51Б-51Г). На Фиг. 51Б показан рост опухоли для каждой отдельной мыши в группе носителя, на Фиг. 51 В - мышей обработанных CD19-CD28 (вариант 15), и на Фиг. 51Г -мышей обработанных CD19-CD28 (вариант 8). Можно видеть, что CD19-CD28 (вариант 8), как единственный агент, индуцировал более сильное ингибирование роста опухоли по сравнению с CD19-CD28 (вариант 15).
ПОДРОБНОЕ ОПИСАНИЕ СУЩНОСТИ ИЗОБРЕТЕНИЯ
Определения
Если не указано иное, все технические и научные термины, используемые в данном документе, имеют значение, обычно используемое в области, к которой принадлежит это изобретение. В целях интерпретации этого описания применимы нижеприведенные определения, и в тех случаях, когда это целесообразно, термины, используемые в единственном числе, также включают множественное число и наоборот.
В контексте данного документа термин «антигенсвязывающая молекула» относится в своем самом широком смысле к молекуле, которая специфически связывает антигенную детерминанту. Примерами антигенсвязывающих молекул являются антитела, мультиспецифические антитела (например, биспецифические антитела), фрагменты антител и каркасные антигенсвязывающие белки.
В контексте данного документа термин «антигенсвязывающий домен, который связывается с опухолеассоциированным антигеном» или «фрагмент, способный к специфическому связыванию с опухолеассоциированным антигеном» относится к полипептидной молекуле, которая специфически связывается с антигенной детерминантой. В одном аспекте антигенсвязывающий домен способен активировать сигнализацию через антиген целевой клетки. В конкретном аспекте антигенсвязывающий домен способен направлять соединение, к которому он присоединен (например, антитело к CD28) к целевому сайту, например, к конкретному типу опухолевой клетки или опухолевой стромы, несущей антигенную детерминанту. Антигенсвязывающие домены, способные к специфическому связыванию с антигеном целевой клетки, включают антитела и их фрагменты, которые определены далее в данном документе. Кроме того, антигенсвязывающие домены, способные к специфическому связыванию с антигеном целевой клетки, включают каркасные антигенсвязывающие белки, которые определены далее в данном документе, в основе которых лежат сконструированные повторяющиеся белки или сконструированные повторяющиеся домены (смотрите, например, WO 2002/020565).
В отношении антигенсвязывающей молекулы, т.е. антитела или его фрагмента термин «антигенсвязывающий домен, который связывается с антигеном клетки-мишени» относится к молекуле, которая содержит участок, который специфически связывается и является комплементарной с частью или со всем антигеном. Антигенсвязывающий домен, способный к специфическому связыванию антигена, может быть обеспечен, например, одним или более вариабельными доменами антитела (также называемыми вариабельными областями антитела). В частности, антигенсвязывающий домен, способный к специфическому связыванию антигена, содержит вариабельную область легкой цепи антитела (VL) и вариабельную область тяжелой цепи антитела (VH). В другом аспекте «антигенсвязывающий домен, способный к специфическому связыванию с опухолеассоциированным антигеном» также может представлять собой Fab-фрагмент или Kpocc-Fab-фрагмент. В другом аспекте «антигенсвязывающий домен, способный к специфическому связыванию с опухолеассоциированным антигеном» также может представлять собой Fab-фрагмент или Kpocc-Fab-фрагмент. В контексте данного документа термины «первый», «второй» или «третий» в отношении антигенсвязывающих доменов и т.д. используются для удобства различения, когда присутствует более одного из каждого типа фрагментов. Использование этих терминов не подразумевает конкретные порядок или ориентацию фрагментов, если явно не указано иное.
В контексте данного документа термин «антигенсвязывающий домен, который связывается с поверхностным антигеном В-клеток» или «фрагмент, способный к специфическому связыванию с поверхностным антигеном В-клеток» относится к полипептидной молекуле, которая специфически связывается с антигенной детерминантой на поверхности В-клеток. В одном аспекте антигенсвязывающий домен способен активировать сигнализацию через антиген целевой клетки. В конкретном аспекте антигенсвязывающий домен способен направлять соединение, к которому он присоединен (например, агониста CD28) к целевому сайту, например, на В-клетке. Антигенсвязывающие домены, способные к специфическому связыванию с поверхностным антигеном В-клеток, включают антитела и их фрагменты, которые определены далее в данном документе. Кроме того, антигенсвязывающие домены, способные к специфическому связыванию с поверхностным антигеном В-клеток, включают каркасные антигенсвязывающие белки, которые определены далее в данном документе, в основе которых лежат сконструированные повторяющиеся белки или сконструированные повторяющиеся домены (смотрите, например, WO 2002/020565).
В контексте данного документа термин «антигенсвязывающий домен, который связывается с антигеном клеточной поверхности множественной миеломы (ММ)» или «фрагмент, способный к специфическому связыванию с антигеном клеточной поверхности множественной миеломы (ММ)» относится к полипептидной молекуле, которая специфически связывается с антигенной детерминантой на клетке множественной миеломы (ММ). В одном аспекте антигенсвязывающий домен способен активировать сигнализацию через антиген целевой клетки. В конкретном аспекте антигенсвязывающий домен способен направлять соединение, к которому он присоединен (например, агониста CD28) к целевому сайту, например, на клетке ММ. Антигенсвязывающие домены, способные к специфическому связыванию с антигеном клеточной поверхности множественной миеломы (ММ), включают антитела и их фрагменты, которые определены далее в данном документе. Кроме того, антигенсвязывающие домены, способные к специфическому связыванию с поверхностным антигеном В-клеток, включают каркасные антигенсвязывающие белки, которые определены далее в данном документе, в основе которых лежат сконструированные повторяющиеся белки или сконструированные повторяющиеся домены (смотрите, например. WO 2002/020565).
Термин «антитело» в данном документе используется в самом широком смысле и охватывает различные структуры антител, включая, но не ограничиваясь этим, моноклональные антитела, поликлональные антитела, моноспецифические и мультиспецифические антитела (например, биспецифические антитела) и фрагменты антител, при условии, что они демонстрируют необходимую антигенсвязывающую активность.
В контексте данного документа термин «моноклональное антитело» относится к антителу, полученному из популяции практически гомогенных антител, т.е. отдельные антитела, составляющие популяцию, идентичны и/или связываются с одним и тем же эпитопом, за исключением возможных вариантных антител, например, содержащих мутации природного происхождения или возникающие во время получения препарата моноклональных антител, при этом такие варианты в целом присутствуют в незначительных количествах. В отличие от препаратов поликлональных антител, которые обычно содержат разные антитела, направленные против разных детерминант (эпитопов), каждое моноклональное антитело препарата моноклональных антител направлено против одной детерминанты на антигене.
В контексте данного документа термин «моноспецифическое антитело» обозначает антитело, которое имеет один или более сайтов связывания, каждый из которых связывается с одним и тем же эпитопом одного и того же антигена. Термин «биспецифический» означает, что антигенсвязывающая молекула способна к специфическому связыванию с по меньшей мере двумя разными антигенными детерминантами. Как правило, биспецифическая антигенсвязывающая молекула содержит два антигенсвязывающих сайта, каждый из которых является специфическим в отношении отличной антигенной детерминанты. При этом биспецифическая антигенсвязывающая молекула также может содержать дополнительные антигенсвязывающие сайты, которые связываются с дополнительными антигенными детерминантами. В определенных аспектах биспецифическая антигенсвязывающая молекула способна одновременно связывать две антигенные детерминанты, в частности, две антигенные детерминанты, экспрессируемые на двух разных клетках или на одной клетке. Термин «биспецифическая» в соответствии с настоящим изобретением также может включать триспецифическую молекулу, содержащую антитело к CD28 и два антигенсвязывающих домена, направленных на два разных антигена клетки-мишени.
Термин «валентный» в контексте представленной заявки обозначает наличие конкретного числа сайтов связывания в антигенсвязывающей молекуле, специфических в отношении одной отличной антигенной детерминанты. Таким образом, термины «двухвалентный», «четырехвалентный» и «шестивалентный» обозначают наличие в антигенсвязывающей молекуле двух сайтов связывания, четырех сайтов связывания и шести сайтов связывания, специфических в отношении определенной антигенной детерминанты, соответственно. В конкретных аспектах изобретения биспецифические антигенсвязывающие молекулы в соответствии с изобретением могут быть одновалентными в отношении определенной антигенной детерминанты, что означает, что они имеют только один сайт связывания для указанной антигенной детерминанты, или же они могут быть двухвалентными или четырехвалентными в отношении определенной антигенной детерминанты, что означает, что они имеют два сайта связывания или четыре сайта связывания, соответственно, для указанной антигенной детерминанты.
В контексте данного документа термины «полноразмерное антитело», «интактное антитело» и «цельное антитело» используются взаимозаменяемо для обозначения антитела, имеющего структуру, практически аналогичную структуре нативного антитела. «Нативные антитела» относятся к молекулам иммуноглобулинов природного происхождения с различной структурой. Например, нативные антитела класса IgG представляют собой гетеротетрамерные гликопротеины массой около 150000 дальтон, состоящие из двух легких цепей и двух тяжелых цепей, связанных дисульфидными связями. В направлении от N- к С-концу каждая тяжелая цепь содержит вариабельную область (VH), также называемую вариабельным тяжелым доменом или вариабельным доменом тяжелой цепи, за которой следуют три константных домена (СН1, СН2 и СН3), также называемые константной областью тяжелой цепи. Аналогично, в направлении от N- к С-концу каждая легкая цепь содержит вариабельную область (VL), также называемую вариабельным легким доменом или вариабельным доменом легкой цепи, за которой следует константный легкий домен (CL), также называемый константной областью легкой цепи. Тяжелая цепь антитела может быть отнесена к одному из пяти типов, называемых α (IgA), δ (IgD), ε (IgE), γ (IgG) или μ (IgM), некоторые из которых могут быть дополнительно разделены на подтипы, например, γ1 (IgGI), γ2 (IgG2), γ3 (IgG3), γ4 (IgG4), α1 (IgA1) и α2 (IgA2). Легкая цепь антитела может быть отнесена к одному из двух типов, называемых каппа (κ) и лямбда (λ), на основании аминокислотной последовательности своего константного домена.
«Фрагмент антитела» относится к молекуле, отличной от интактного антитела, которая содержит часть интактного антитела, которая связывает антиген, с которым связывается интактное антитело. Примеры фрагментов антител включают, но не ограничиваются этим, Fv, Fab, Fab', Fab'-SH, F(ab')2; диатела, триатела, тетратела, кросс-Fab-фрагменты; линейные антитела; молекулы одноцепочечных антител (например, scFv); и однодоменные антитела. Обзор некоторых фрагментов антител смотрите в Hudson et al., Nat Med 9, 129-134 (2003). Обзор фрагментов scFv смотрите, например, в  , в The Pharmacology of Monoclonal Antibodies, vol. 113, Rosenburg and Moore eds., Springer-Verlag, New York, pp.269-315 (1994); также смотрите WO 93/16185; и патенты США №№5571894 и 5587458. Обсуждение фрагментов Fab и F(ab')2, содержащих остатки эпитопа связывания рецептора реутилизации и имеющих увеличенное время полужизни in vivo, смотрите в патенте США №5869046. Диатела представляют собой фрагменты антител с двумя антигенсвязывающими сайтами, которые могут быть двухвалентными или биспецифическими, смотрите, например, ЕР 404097; WO 1993/01161; Hudson et al., Nat Med 9, 129-134 (2003); и Hollinger et al., Proc Nail Acad Sci USA 90, 6444-6448 (1993). Триатела и тетратела также описаны в Hudson et al., Nat Med 9, 129-134 (2003). Однодоменные антитела представляют собой фрагменты антител, содержащие весь или часть вариабельного домена тяжелой цепи или весь или часть вариабельного домена легкой цепи антитела. В определенных вариантах осуществления однодоменное антитело представляет собой человеческое однодоменное антитело (Domantis, Inc., Waltham, MA; смотрите, например, патент США №6248516 В1). Фрагменты антител можно получать различными способами, включая, но не ограничиваясь этим, протеолитическое расщепление интактного антитела, а также выработку рекомбинантными клетками-хозяевами (например, Е. coli или фага), как описано в данном документе.
, в The Pharmacology of Monoclonal Antibodies, vol. 113, Rosenburg and Moore eds., Springer-Verlag, New York, pp.269-315 (1994); также смотрите WO 93/16185; и патенты США №№5571894 и 5587458. Обсуждение фрагментов Fab и F(ab')2, содержащих остатки эпитопа связывания рецептора реутилизации и имеющих увеличенное время полужизни in vivo, смотрите в патенте США №5869046. Диатела представляют собой фрагменты антител с двумя антигенсвязывающими сайтами, которые могут быть двухвалентными или биспецифическими, смотрите, например, ЕР 404097; WO 1993/01161; Hudson et al., Nat Med 9, 129-134 (2003); и Hollinger et al., Proc Nail Acad Sci USA 90, 6444-6448 (1993). Триатела и тетратела также описаны в Hudson et al., Nat Med 9, 129-134 (2003). Однодоменные антитела представляют собой фрагменты антител, содержащие весь или часть вариабельного домена тяжелой цепи или весь или часть вариабельного домена легкой цепи антитела. В определенных вариантах осуществления однодоменное антитело представляет собой человеческое однодоменное антитело (Domantis, Inc., Waltham, MA; смотрите, например, патент США №6248516 В1). Фрагменты антител можно получать различными способами, включая, но не ограничиваясь этим, протеолитическое расщепление интактного антитела, а также выработку рекомбинантными клетками-хозяевами (например, Е. coli или фага), как описано в данном документе.
Расщепление интактных антител папаином позволяет получить два идентичных антигенсвязывающих фрагмента, называемых «Fab»-фрагментами, каждый из которых содержит вариабельные домены тяжелой и легкой цепи и также константный домен легкой цепи и первый константный домен (СН1) тяжелой цепи. Таким образом, в контексте данного документа термин «Fab-фрагмент» относится к фрагменту антитела, содержащему фрагмент легкой цепи, содержащий вариабельный домен легкой цепи (VL) и константный домен легкой цепи (CL), и вариабельный домен тяжелой цепи (VH) и первый константный домен (СН1) тяжелой цепи. Fab'-фрагменты отличаются от Fab-фрагментов добавлением нескольких остатков в карбокси-конце CH1-домена тяжелой цепи, включая один или более остатков цистеина из шарнирной области антитела. Fab'-SH представляют собой Fab'-фрагменты, в которых остаток(ки) цистеина константных доменов несет(ут) свободную тиольную группу. Обработка пепсином приводит к получению F(ab')2-фрагмента, который имеет два антигенсвязывающих сайта (два Fab-фрагмента) и часть Fc-области.
Термин «кросс-Fab-фрагмент», или «xFab-фрагмент», или «кроссоверный Fab-фрагмент» относится к Fab-фрагменту, в котором вариабельные области или константные области тяжелой и легкой цепи поменяны. В биспецифических антителах по изобретению возможны и содержатся два разных варианта композиции цепи кроссоверной Fab-молекулы:
С одной стороны поменяны вариабельные области тяжелой и легкой цепи Fab, т.е. кроссоверная Fab-молекула содержит пептидную цепь, состоящую из вариабельного домена легкой цепи (VL) и константного домена тяжелой цепи (СН1), и пептидную цепь, состоящую из вариабельного домена тяжелой цепи (VH) и константного домена легкой цепи (CL). Эта кроссоверная Fab-молекула также называется кросс-Fab (VLVH). С другой стороны, когда поменяны константные области тяжелой и легкой цепи Fab, кроссоверная Fab-молекула содержит пептидную цепь, состоящую из вариабельного домена тяжелой цепи (VH) и константного домена легкой цепи (CL), и пептидную цепь, состоящую из вариабельного домена легкой цепи (VL) и константного домена тяжелой цепи (СН1). Эта кроссоверная Fab-молекула также называется кросс-Fab (CLCHI).
«Одноцепочечный Fab-фрагмент» или «scFab» представляет собой полипептид, состоящий из вариабельного домена тяжелой цепи антитела (VH), константного домена антитела 1 (СН1), вариабельного домена легкой цепи антитела (VL), константного домена легкой цепи антитела (CL) и линкера, причем указанные домены антитела и указанный линкер имеют один из следующих вариантов упорядочения в направлении от N-конца к С-концу: а) VH-CH1-линкер-VL-CL, б) VL-CL-линкер-VH-CH1, в) VH-CL-линкер-VL-СН1 или г) VL-CH1-линкер-VH-CL; и причем указанный линкер представляет собой полипептид из по меньшей мере 30 аминокислот, предпочтительно от 32 до 50 аминокислот. Указанные одноцепочечные Fab-фрагменты стабилизированы посредством естественной дисульфидной связи между C1-доменом и СН1-доменом. Кроме того, эти одноцепочечные Fab-молекулы можно дополнительно стабилизировать путем создания межцепочечных дисульфидных связей посредством вставки остатков цистеина (например, в позиции 44 в вариабельной области тяжелой цепи и позиции 100 в вариабельной области легкой цепи в соответствии с нумерацией Kabat).
«Кроссоверный одноцепочечный Fab-фрагмент» или «x-scFab» представляет собой полипептид, состоящий из вариабельного домена тяжелой цепи антитела (VH), константного домена антитела 1 (СН1), вариабельного домена легкой цепи антитела (VL), константного домена легкой цепи антитела (CL) и линкера, причем указанные домены антитела и указанный линкер имеют один из следующих вариантов упорядочения в направлении от N-конца к С-концу: a) VH-CL-линкер-VL-CH1 и б) VL-CH1-линкер-VH-CL; где VH и VL вместе образуют антигенсвязывающий сайт, который специфически связывается с антигеном, и при этом указанный линкер представляет собой полипептид из по меньшей мере 30 аминокислот. Кроме того, эти x-scFab-молекулы можно дополнительно стабилизировать путем создания межцепочечных дисульфидных связей посредством вставки остатков цистеина (например, в позиции 44 в вариабельной области тяжелой цепи и позиции 100 в вариабельной области легкой цепи в соответствии с нумерацией Kabat).
«Одноцепочечный вариабельный фрагмент (scFv)» представляет собой слитый белок из вариабельных областей тяжелой (VH) и легкой цепей (VL) антитела, соединенный коротким линкерным пептидом из от десяти до около 25 аминокислот. Линкер обычно обогащен глицином для гибкости, а также серином или треонином для растворимости, и может соединять N-конец VH с С-концом VL или наоборот. Этот белок сохраняет специфичность исходного антитела несмотря на удаление константных областей и внесение линкера. scFv-антитела описаны, например, в Houston, J.S., Methods in Enzymol. 203 (1991) 46-96). Кроме того, фрагменты антител содержат одноцепочечные полипептиды, имеющие характеристики VH-домена, а именно способность к сборке с VL-доменом, или VL-домена, а именно способность к сборке с VH-доменом в функциональный антигенсвязывающий сайт и, таким образом, обеспечивающие антигенсвязывающие свойства полноразмерного полипептида.
«Каркасные антигенсвязывающие белки» известны в данной области техники, например, фибронектин и сконструированные белки с анкириновыми повторами (дарпины или DARPin) использовали в качестве альтернативных каркасов для антигенсвязывающих доменов, смотрите, например, Gebauer and Skerra, Engineered protein scaffolds as next-generation antibody therapeutics. Curr Opin Chem Biol 13:245-255 (2009) и Stumpp et al., Darpins: A new generation of protein therapeutics. Drug Discovery Today 13: 695-701 (2008). В одном аспекте изобретения каркасный антигенсвязывающий белок выбран из группы, состоящей из CTLA-4 (эвитело), липокалинов (антикалин), полученных из протеина А молекул, таких как Z-домен протеина А (аффитело), А-домена (авимер/макситело), сывороточного трансферрина (транс-тело);
сконструированного белка с анкириновыми повторами (дарпин), вариабельного домена легкой цепи или тяжелой цепи антитела (однодоменное антитело, sdAb), вариабельного домена тяжелой цепи антитела (нанотело, aVH), фрагментов VNAR, фибронектина (аднектин), лектинового домена С-типа (тетранектин);
вариабельного домена бета-лактамазы нового антигенного рецептора (фрагменты VNAR.). человеческого гамма-кристаллина или убиквитина (молекулы аффилина); домена типа домена Куница ингибиторов протеазы человека, микротел, таких как белки семейства ноттина, пептидные аптамеры и фибронектин (аднектин). CTLA-4 (ассоциированный с цитотоксическими Т-лимфоцитами антиген 4) представляет собой рецептор семейства CD28, экспрессируемый преимущественно на CD4+ Т-клетках. Его внеклеточный домен имеет укладку, подобную вариабельному домену Ig. Петли, соответствующие CDR антител, могут быть замещены гетерологичной последовательностью для придания отличных связывающих свойств. Молекулы CTLA-4, сконструированные так, чтобы иметь разные варианты специфичности связывания, также известны как эвитела (например, US7166697B1). Эвитела имеют приблизительно такой же размер, что и выделенная вариабельная область антитела (например, доменного антитела). Дополнительные подробности смотрите в Journal of Immunological Methods 248 (1-2), 31-45 (2001). Липокалины представляют семейство внеклеточных белков, которые переносят небольшие гидрофобные молекулы, такие как стероиды, билины, ретиноиды и липиды. Они имеют жесткую бета-складчатую вторичную структуру с рядом петель в открытом конце конической структуры, которая может быть сконструирована так, чтобы связываться с разными целевыми антигенами. Размер антикалинов составляет 160-180 аминокислот, и они получены из липокалинов. Дополнительные подробности смотрите в Biochim Biophys Acta 1482: 337-350 (2000), US7250297B1 и US20070224633. Аффитело представляет собой каркас, полученный из протеина A Staphylococcus aureus, который может быть сконструирован так, чтобы связывать антиген. Домен состоит из трехспирального пучка приблизительно из 58 аминокислот. Путем рандомизации поверхностных остатков были созданы библиотеки. Дополнительные подробности смотрите в Protein Eng. Des. Sel. 2004, 17, 455-462 и ЕР 1641818А1. Авимеры представляют собой многодоменные белки, полученные из семейства А-доменных каркасов. Нативные домены приблизительно из 35 аминокислот принимают определенную дисульфидно связанную структуру. Разнообразие создают путем перетасовки природной вариации, демонстрируемой семейством А-доменов. Дополнительные подробности смотрите в Nature Biotechnology 23(12), 1556 - 1561 (2005) and Expert Opinion on Investigational Drugs 16(6), 909-917 (June 2007). Трансферрин представляет собой мономерный сывороточный транспортный гликопротеин. Трансферрины могут быть сконструированы так, чтобы связывать разные целевые антигены, путем вставки пептидных последовательностей в пермиссивную поверхностную петлю. Примеры сконструированных трансферриновых каркасов включают транс-тело. Дополнительные подробности смотрите в J. Biol. Chem 274, 24066-24073 (1999). Сконструированные белки с анкириновыми повторами (дарпины или DARPin) получены из анкирина, который принадлежит семейству белков, которые опосредуют присоединение интегральных мембранных белков к цитоскелету. Один анкириновый повтор представляет собой мотив из 33 остатков, состоящий из двух альфа-спиралей и бета-изгиба. Их можно сконструировать так, чтобы они связывали разные целевые антигены, путем рандомизации остатков в первой альфа-спирали и бета-изгибе каждого повтора. Их поверхность связывания можно увеличить за счет увеличения числа модулей (способ созревания аффинности). Дополнительные подробности смотрите в J. Mol. Biol. 332, 489-503 (2003), PNAS 100(4), 1700-1705 (2003) и J. Mol. Biol. 369, 1015-1028 (2007) и US20040132028A1. Однодоменное антитело представляет собой фрагмент антитела, состоящий из одного мономерного вариабельного домена антитела. Первые однодоменные антитела были получены из вариабельного домена тяжелой цепи антитела верблюжьих (нанотела или фрагменты VHН). Кроме того, термин однодоменное антитело включает автономный человеческий вариабельный домен тяжелой цепи (aVH) или фрагменты VNAR. полученные от акул. Фибронектин представляет собой каркас, который может быть сконструирован так, чтобы связывать антиген. Аднектины состоят из остова природной аминокислотной последовательности 10-го домена из 15 повторяющихся единиц человеческого фибронектина типа III (FN3). Три петли в одном конце бета-сандвича могут быть сконструированы так, чтобы аднектин мог специфически распознавать представляющую интерес терапевтическую мишень. Дополнительные подробности смотрите в Protein Eng. Des. Sel. 18, 435-444 (2005), US20080139791, W02005056764 и US6818418B1. Пептидные аптамеры представляют собой комбинаторные молекулы распознавания, которые состоят из константного каркасного белка, как правило, тиоредоксина (TrxA), которые содержит пространственно-ограниченную вариабельную пептидную петлю, вставленную в активном сайте. Дополнительные подробности смотрите в Expert Opin. Biol. Ther. 5, 783-797 (2005). Микротела получены из микропротеинов природного происхождения длиной 25 50 аминокислот, которые содержат 34 цистеиновых мостика; примеры микропротеинов включают KalataBI, конотоксин и ноттины. Микропротеины имеют петлю, которая может быть сконструирована так, чтобы содержать до 25 аминокислот, не влияя на общую укладку белка. Дополнительные подробности в отношении сконструированных доменов ноттинов смотрите в W02008098796.
«Антигенсвязывающая молекула, которая связывается с тем же эпитопом», что и референсная молекула, относится к антигенсвязывающей молекуле, которая в конкурентном анализе блокирует связывание референсной молекулы с ее антигеном на 50% или более, и наоборот, референсная молекула в конкурентном анализе блокирует связывание антигенсвязывающей молекулы с ее антигеном на 50% и более.
Термин «антигенсвязывающий домен» относится к части антигенсвязывающей молекулы, которая содержит участок, который специфически связывается и является комплементарным с частью или со всем антигеном. Если антиген является крупным, антигенсвязывающая молекула может связываться только с конкретной частью антигена, и эта часть называется эпитопом. Антигенсвязывающий домен можно обеспечить, например, одним или более вариабельными доменами (также называемыми вариабельными областями). Предпочтительно антигенсвязывающий домен содержит вариабельный домен легкой цепи антитела (VL) и вариабельный домен тяжелой цепи антитела (VH).
В контексте данного документа «антигенная детерминанта» является синонимом терминам «антиген» и «эпитоп» и относится к сайту (например, непрерывному участку аминокислот или конформационной конфигурации, состоящей из разных областей не являющихся непрерывными аминокислот) на полипептидной макромолекуле, с которым связывается антигенсвязывающий фрагмент с образованием комплекса антигенсвязывающий фрагмент антиген. Применимые антигенные детерминанты можно обнаружить, например, на поверхностях опухолевых клеток, на поверхностях инфицированных вирусом клеток, на поверхностях пораженных заболеванием клеток, на поверхности иммунных клеток, в свободном виде в сыворотке крови и/или во внеклеточном матриксе (ВКМ). Белки, применимые в контексте данного документа, могут представлять собой любую нативную форму белков из любого источника, относящегося к позвоночным, включая млекопитающих, таких как приматы (например, люди) и грызуны (например, мыши и крысы), если не указано иное. В конкретном варианте осуществления антиген представляет собой человеческий белок. Когда в данном документе приводится ссылка на конкретный белок, этот термин включает «полноразмерный» непроцессированный белок, а также любую форму белка, которая является результатом процессинга в клетке. Этот термин также охватывает варианты белка природного происхождения, например, сплайс-варианты или аллельные варианты.
Под выражением «специфическое связывание» подразумевается, что связывание является избирательным в отношении антигена и может быть отделено от нежелательных или неспецифических взаимодействий. Способность антигенсвязывающей антигенсвязывающей молекулы связываться с конкретным антигеном можно определить с помощью ферментного иммуносорбентного анализа (ELISA) или других методик, известных специалисту в данной области техники, например, метода поверхностного плазменного резонанса (ППР) (с анализом на приборе BIAcore) (Liljeblad et al., Glyco J 17, 323-329 (2000)), и традиционных анализов связывания (Heeley, Endocr Res 28, 217-229 (2002)). В одном варианте осуществления степень связывания антигенсвязывающей молекулы с неродственным белком составляет менее чем около 10% связывания антигенсвязывающей молекулы с антигеном при измерении, например, методом ППР. В определенных вариантах осуществления молекула, которая связывается с антигеном, имеет константу диссоциации (Kд) ≤1 мкМ, ≤100 нМ, ≤10 нМ, ≤1 нМ, ≤0,1 нМ, ≤0,01 нМ или ≤0,001 нМ (например, 10-8 М или менее, например, от 10-8 М до 10-13 М, например, от 10-9 М до 10-13 М).
«Аффинность» или «аффинность связывания» относится к силе суммарных нековалентных взаимодействий между одним сайтом связывания молекулы (например, антитела) и ее партнером по связыванию (например, антигеном). Если не указано иное, в контексте данного документа «аффинность связывания» относится к характерной аффинности связывания, которая отображает 1:1 взаимодействие между членами связывающейся пары (например, антителом и антигеном). Аффинность молекулы Х в отношении ее партнера Y в общем случае можно выразить константой диссоциации (Kд), которая представляет собой отношение констант диссоциации и ассоциации (kдисс. и kacc., соответственно). Таким образом, эквивалентные аффинности могут включать разные константы скорости при условии, что отношение между константами скорости остается одинаковым. Аффинность можно измерять с помощью общепринятых методов, известных в данной области техники, включая описанные в данном документе. Конкретным методом для измерения аффинности является поверхностный плазменный резонанс (ППР).
В контексте данного документа «опухолеассоциированный антиген» или ОАА относится к антигенной детерминанте, представленной на поверхности клетки-мишени, например, клетки в опухоли, такой как раковая клетка, клетка опухолевой стромы, злокачественный В-лимфоцит или клетка меланомы. В определенных аспектах антиген клетки-мишени представляет собой антиген на поверхности опухолевой клетки. В одном аспекте ОАА выбран из группы, состоящей из белка активации фибробластов (FAP), карциноэмбрионального антигена (СЕА), рецептора фолиевой кислоты альфа (FolR1), ассоциированного с меланомой хондроитинсульфата протеогликана (MCSP), рецептора эпидермального фактора роста (EGFR), рецептора 2 эпидермального фактора роста человека (HER2), p95HER2, ЕрСАМ, HER3, CD30 или TPBG (5Т4), CD19, CD79b, CD20, CD22, CD37, CD38, ВСМА и GPRC5D. В одном конкретном аспекте ОАА выбран из группы, состоящей из белка активации фибробластов (FAP), карциноэмбрионального антигена (СЕА), рецептора фолиевой кислоты альфа (FolR1), ассоциированного с меланомой хондроитинсульфата протеогликана (MCSP), рецептора эпидермального фактора роста (EGFR), рецептора 2 эпидермального фактора роста человека (HER2) и p95HER2. В другом конкретном аспекте ОАА выбран из группы, состоящей из белка активации фибробластов (FAP), карциноэмбрионального антигена (СЕА), ЕрСАМ, HER3, CD30 или TPBG (5T4). В одном конкретном аспекте опухолеассоциированный антиген представляет собой белок активации фибробластов (FAP) или карциноэмбриональный антиген (СЕА). В одном аспекте ОАА представляет собой поверхностный антиген В-клеток, выбранный из группы, состоящей из CD19, CD79b, CD20, CD22 и CD37, в частности CD19 и CD79b. В одном аспекте ОАА представляет собой антиген клеточной поверхности множественной миеломы (ММ), выбранный из группы, состоящей из CD38, ВСМА и GPRC5D.
Термин «белок активации фибробластов (FAP)», также известный как FAP пролилэндопептидаза или сепраза (ЕС 3.4.21), относится к любому нативному FAP из любого источника, относящегося к позвоночным, включая млекопитающих, таких как приматы (например, люди), отличные от человека приматы (например, яванские макаки) и грызуны (например, мыши и крысы), если не указано иное. Этот термин охватывает «полноразмерный» непроцессированный FAP, а также любую форму FAP, полученную в результате процессинга в клетке. Этот термин также охватывает варианты FAP природного происхождения, например, сплайс-варианты или аллельные варианты. В одном варианте осуществления антигенсвязывающая молекула по изобретению способна к специфическому связыванию с FAP человека, мыши и/или яванского макака. Аминокислотная последовательность FAP человека приведена под номером доступа UniProt (www.uniprot.org) Q12884 (версия 149, SEQ ID NO:2) или NCBI (www.ncbi.nlm.nih.gov/) RefSeq NP_004451.2. Внеклеточный домен (ВКД) FAP человека простирается от аминокислотной позиции 26 до 760.
Аминокислотная последовательность ВКД His-меченного FAP человека приведена в SEQ ID NO:135. Аминокислотная последовательность FAP мыши приведена под номером доступа Р97321 (версия 126, SEQ ID NO:136) или NCBI RefSeq NP_032012.1. Внеклеточный домен (ВКД) FAP мыши простирается от аминокислотной позиции 26 до 761. SEQ ID NO:137 иллюстрирует аминокислотную последовательность ВКД His-меченного FAP мыши. SEQ ID NO:138 иллюстрирует аминокислотную последовательность ВКД His-меченного FAP яванского макака. Предпочтительно анти-FAP связывающая молекула по изобретению связывается с внеклеточным доменом FAP.
Термин «карциноэмбриональный антиген (СЕА)», также известный как родственная карциноэмбриональному антигену молекула адгезии 5 (СЕАСАМ5), относится к любому нативному СЕА из любого источника, относящегося к позвоночным, включая млекопитающих, таких как приматы (например, люди), отличные от человека приматы (например, яванские макаки) и грызуны (например, мыши и крысы), если не указано иное. Аминокислотная последовательность СЕА человека приведена под номером доступа UniProt №Р06731 (версия 151, SEQ ID NO:3). СЕА давно был идентифицирован как опухолеассоциированный антиген (Gold and Freedman, J Exp Med., 121:439-462, 1965; Berinstein N. L., J Clin Oncol., 20:2197-2207, 2002). Изначально классифицированный как белок, экспрессируемый только в фетальной ткани, сейчас СЕА был обнаружен в нескольких нормальных взрослых тканях. Эти ткани в основном являются эпителиальными по происхождению, включая клетки желудочно-кишечного, дыхательного и мочеполового тракта, а также клетки толстой кишки, шейки матки, потовых желез и предстательной железы (Nap et al., Tumour Biol., 9(2-3): 145-53, 1988; Nap et al., Cancer Res., 52(8):2329-23339, 1992). Опухоли эпителиального происхождения, а также их метастазы, содержат СЕА как опухолеассоциированный антиген. Хотя само по себе наличие СЕА не свидетельствует о трансформации в раковую клетку, распределение СЕА является показательным. В нормальной ткани СЕА в общем случае экспрессируется на апикальной поверхности клетки (Hammarstrom S., Semin Cancer Biol. 9(2):67-81 (1999)), делая ее недоступной для антитела в кровотоке. В отличие от нормальной ткани, существует тенденция к экспрессии СЕА по всей поверхности раковых клеток (Hammarstrom S., Semin Cancer Biol. 9(2):67-81 (1999)). Это изменение профиля экспрессии делает СЕА доступным для связывания антителом в раковых клетках. Помимо этого, в раковых клетках экспрессия СЕА повышается. Кроме того, повышенная экспрессия СЕА способствует повышению внутриклеточной адгезии, что может привести к метастазам (Marshall J., Semin Oncol., 30(a Suppl. 8):30-6, 2003). Распространенность экспрессии СЕА в различных опухолевых элементах в общем случае является очень высокой. В соответствии с опубликованными данными собственный анализ, проведенный на тканевых образцах, подтвердил его высокую распространенность, составляющую приблизительно 95% при колоректальной карциноме (КРК), 90% при раке поджелудочной железы, 80% при раке желудка, 60% при немелкоклеточном раке легкого (НМРЛ, при котором он коэкспрессируется с HER3) и 40% при раке молочной железы; низкая экспрессия была обнаружена при мелкоклеточном раке легкого и глиобластоме.
СЕА легко отщепляется от поверхности клетки и попадает в кровоток из опухолей, как напрямую, так и через лимфатическую систему. Из-за этого свойства уровень сывороточного СЕА использовали как клинический маркер для диагностики раков и скрининга в отношении повторного появления раков, в частности колоректального рака (Goldenberg D M., The International Journal of Biological Markers, 7:183-188, 1992; Chau I., et al., J Clin Oncol., 22:1420-1429, 2004; Flamini et al., Clin Cancer Res; 12(23):6985-6988, 2006).
Термин «молекула адгезии эпителиальных клеток (ЕрСАМ)» относится к любому нативному ЕрСАМ из любого источника, относящегося к позвоночным, включая млекопитающих, таких как приматы (например, люди), отличные от человека приматы (например, яванские макаки) и грызуны (например, мыши и крысы), если не указано иное. Этот термин охватывает «полноразмерный» непроцессированный ЕрСАМ, а также любую форму ЕрСАМ, полученную в результате процессинга в клетке. Этот термин также охватывает варианты ЕрСАМ природного происхождения, например, сплайс-варианты или аллельные варианты. В одном варианте осуществления антигенсвязывающая молекула по изобретению способна к специфическому связыванию с ЕрСАМ человека, мыши и/или яванского макака. Аминокислотная последовательность ЕрСАМ человека приведена под номером доступа UniProt (www.uniprot.org) Р16422 (версия 167, SEQ ID NO:68) или NCBI (www.ncbi.nlm.nih.gov/) RefSeq NP_002345.2. Аминокислотная последовательность ЕрСАМ мыши приведена под номером доступа UniProt (www.uniprot.org) Q99JW5 (версия 111, SEQ ID NO:75) или NCBI (www.ncbi.nlm.nih.gov/) RefSeq NP 032558.2. Молекула адгезии эпителиальных клеток (ЕрСАМ), также известная как трансдуктор кальциевого сигнала 1 (TACSTD1), 17-1А и CD326, представляет собой ~ 40 кДа трансмембранный гликопротеин I типа, который часто сверхэкспрессируется в раках эпителиального происхождения и раковыми стволовыми клетками и, следовательно, является молекулой, представляющей значительный интерес для терапии и диагностики. Внеклеточный домен ЕрСАМ может отщепляться с образованием растворимой внеклеточной молекулы ЕрЕХ и внутриклеточной молекулы EpICD. Было показано, что EpICD связывается с другими белками с образованием ядерного комплекса, который оказывает повышающую регуляцию на экспрессию геном, способствующих пролиферации клеток. ЕрСАМ также может быть вовлечен в перенос мезенхимальных клеток (ЕМТ) и может способствовать образованию крупных метастазов.
«CD30» или «TNFRSF8» является членом суперсемейства рецепторов фактора некроза опухолей. Его экспрессия характерна для определенных гемопоэтических злокачественных образований, включая, помимо прочего, анапластическую крупноклеточную лимфому и лимфому Ходжкина. Вариабельная экспрессия CD30 как на нормальных, так и на злокачественных лимфоидных клетках заставила сфокусировать исследовательскую работу на понимании патогенеза повышенной регуляции CD30, его вклада в лимфомагенез за счет анти-апоптотических механизмов и его эффекта на выживаемость клеток. Учитывая ограничение CD30 определенными типами опухолей, логическим продолжением этого была бы попытка применять его в качестве терапевтического агента. CD30 представляет собой 120 кДа трансмембранный гликопротеиновый рецептор, принадлежащий к суперсемейству рецепторов фактора некроза опухолей (TNFR), а внутриклеточный, трансмембранный и внеклеточный домены и аминокислотная последовательность CD30 человека приведены под номером доступа UniProt №Р28908 (SEQ ID NO:472).
Термин «TPBG» относится к трофобластному гликопротеину, также называемому «5Т4». TBPG представляет собой богатый лейцином трансмембранный гликопротеин, участвующий в клеточной адгезии. У взрослых этот белок экспрессируется на высоких уровнях во многих опухолевых тканях и связан с неблагоприятным клиническим результатом при многих видах рака. Он относится к любому нативному TPBG из любого источника, относящегося к позвоночным, включая млекопитающих, таких как приматы (например, люди), отличные от человека приматы (например, яванские макаки) и грызуны (например, мыши и крысы), если не указано иное. Аминокислотная последовательность TPBG человека приведена под номером доступа UniProt №Q13641 (SEQ ID NO:473).
Термин «FolR1» относится к рецептору фолиевой кислоты альфа и был идентифицирован как потенциальная прогностическая и терапевтическая мишень для ряда видов рака. Он относится к любому нативному FolR1 из любого источника, относящегося к позвоночным, включая млекопитающих, таких как приматы (например, люди), отличные от человека приматы (например, яванские макаки) и грызуны (например, мыши и крысы), если не указано иное. Аминокислотная последовательность FolRl человека приведена под номером доступа UniProt №Р 15328 (SEQ ID NO: 139), мышиный FolR1 имеет аминокислотную последовательность согласно номеру доступа UniProt №Р35846 (SEQ ID NO:140), а FolR1 яванского макака имеет аминокислотную последовательность, приведенную под номером доступа UniProt №G7PR14 (SEQ ID NO:141). FolR1 представляет собой N-гликозилированный белок, экспрессируемый на плазматической мембране клеток. FolR1 имеет высокую аффинность в отношении фолиевой кислоты и в отношении нескольких производных фолиевой кислоты и опосредует доставку физиологического фолата, 5-метилтетрагидрофолата, в клетки. FOLR1 является желательной мишенью для FOLR1-направленной терапии рака, поскольку он сверхэкспрессируется в подавляющем большинстве видов рака яичников, а также многих видах рака матки, эндометрия, поджелудочной железы, почек, легких и молочной железы, тогда как экспрессия FOLR1 в нормальных тканях ограничена апикальной мембраной эпителиальных клеток в проксимальных канальцах почки, альвеолярных пневмоцитах легкого, мочевом пузыре, семенниках, хориоидном сплетении и щитовидной железе. В недавних исследованиях определили, что экспрессия FolR1 является исключительно высокой при трижды негативных раках молочной железы (Necela et al. PloS One 2015, 10(3), e0127133).
Термин «ассоциированный с меланомой хондроитинсульфата протеогликан (MCSP)», также известный как хондроитинсульфата протеогликан 4 (CSPG4), относится к любому нативному MCSP из любого источника, относящегося к позвоночным, включая млекопитающих, таких как приматы (например, люди), отличные от человека приматы (например, яванские макаки) и грызуны (например, мыши и крысы), если не указано иное. Аминокислотная последовательность MCSP человека приведена под номером доступа UniProt №Q6UVK1 (версия 103, SEQ ID NO:142). MCSP представляет собой высокогликозилированный интегральный мембранный хондроитинсульфата протеогликан, состоящий из N-связанного 280 кДа компонента гликопротеина и 450 кДа компонента хондроитинсульфата протеогликана, экспрессируемого на клеточной мембране (Ross et al., Arch. Biochem. Biophys. 1983, 225:370-38). MCSP более широко распределен в ряде нормальных и трансформированных клеток. В частности, MCSP встречается практически во всех базальных клетках эпидермиса. MCSP дифференциально экспрессируется в клетках меланомы, и было обнаружено, что он экспрессируется в более чем 90% проанализированных доброкачественных невусах и меланомных поражениях. Также было обнаружено, что MCSP экспрессируется в опухолях немеланоцитарного происхождения, включая базальноклеточную карциному, различные опухоли нервного гребня и карциномы молочной железы.
Термин «рецептор эпидермального фактора роста (EGFR)», также называемый протоонкогеном с-ErbB-1 или рецепторной тирозин-протеинкиназой erbB-1, относится к любому нативному EGFR из любого источника, относящегося к позвоночным, включая млекопитающих, таких как приматы (например, люди), отличные от человека приматы (например, яванские макаки) и грызуны (например, мыши и крысы), если не указано иное. Аминокислотная последовательность EGFR человека приведена под номером доступа UniProt №Р00533 (версия 211, SEQ ID NO:143). Протоонкоген «HER2», (рецептор эпидермального фактора роста 2 человека) кодирует протеинтирозинкиназу (p185HER2), которая является родственной и в некоторой степени гомологичной рецептору эпидермального фактора роста человека. HER2 также известен в данной области техники как с-erbB-2 и иногда под названием крысиный гомолог, пей. Амплификация и/или сверхэкспрессия HER2 ассоциируется с многими злокачественными образованиями у человека и, по-видимому, полностью вовлечена в прогрессирование 25-30% раков молочной железы и яичников человека. Кроме того, степень амплификации обратно коррелирует с наблюдаемым медианным временем выживаемости пациентов (Slamon, D. J. et al., Science 244:707-712 (1989)). Аминокислотная последовательность HER2 человека приведена под номером доступа UniProt №Р04626 (версия 230, SEQ ID NO:144). В контексте данного документа термин «p95HER2» относится к карбокси-концевому фрагменту (CTF) белка рецептора HER2, который также известен как «611-CTF» или «100-115 кДа p95HER2». Фрагмент p95HER2 создается в клетке посредством инициации трансляции мРНК HER2 в позиции кодона 611 полноразмерной молекулы HER2 (Anido et al, EMBO J 25; 3234-44 (2006)). Он имеет молекулярную массу от 100 до 115 кДа и экспрессируется на клеточной мембране, где он может образовывать гомодимеры, поддерживаемые межмолекулярными дисульфидными связями (Pedersen et al., Mol Cell Biol 29, 3319-31 (2009)). Типовая последовательность p95HER2 приведена в SEQ ID NO:145.
«HER3» или «ErbB3» (рецептор эпидермального фактора роста 3 человека), как и другие члены семейства рецепторных тирозинкиназ ErbB, состоит из внеклеточного домена, трансмембранного домена и внутриклеточного домена. Внеклеточный домен состоит из четырех субдоменов (I-IV). Субдомены I и III обогащены лейцином и главным образом участвуют в связывании лиганда. Субдомены II и IV обогащены цистеином и наиболее вероятно вносят свой вклад в конформацию и стабильность белка посредством образования дисульфидных связей. Субдомен II также содержит петлю димеризации, необходимую для образования димера. Цитоплазматический домен содержит околомембранный сегмент, киназный домен и С-концевой домен. Хотя доказательства того, что сверхэкспрессия, конститутивная активация или только мутация ErbB3 являются онкогенными, отсутствуют, https://en.wikipedia.org/wiki/ERBB3 - cite_note-pmid8632008-18 этот белок, как партнер по гетеродимеризации, что наиболее важно, с ErbB2, вовлечен в рост, пролиферацию, химиотерапевтическую резистентность и стимуляцию инвазии и метастазирования. ErbB3 связан с резистентностью к нацеленным терапевтическим средства при многих видах рака. Аминокислотная последовательность HER3 человека приведена под номером доступа UniProt №Р21860 (версия 224, SEQ ID NO:471)
В контексте данного документа «поверхностный антиген В-клеток» относится к антигенной детерминанте, представленной на поверхности В-лимфоцита, в частности злокачественного В-лимфоцита (в этом случае антиген также называется «злокачественным поверхностным антигеном В-клеток»). Несколько поверхностных антигенов В-клеток интересны в терминах иммунотерапии гематологических злокачественных новообразований. В одном аспекте поверхностный антиген В-клеток выбран из группы, состоящей из CD 19, CD79b, CD20, CD22 и CD37.
Термин «CD19» относится к В-лимфоцитарному антигену CD 19, также известному как В-лимфоцитарный поверхностный антиген В4 или Т-клеточный поверхностный антиген Leu-12, и включает любой нативный CD19 из любого источника, относящегося к позвоночным, включая млекопитающих, таких как приматы (например, люди), отличные от человека приматы (например, яванские макаки) и грызуны (например, мыши и крысы), если не указано иное. Аминокислотная последовательность CD19 человека приведена под номером доступа UniProt №Р15391 (версия 160, SEQ ID NO:434). Этот термин охватывает «полноразмерный» непроцессированный CD19 человека, а также любую форму CD19 человека, полученную в результате процессинга в клетке, при условии, что описанное в данном документе антитело связывается с ней. CD19 является структурно отличным рецептором клеточной поверхности, экспрессируемым на поверхности В-клеток человека, включая, но не ограничиваясь этим, пре-В-клетки, В-клетки на ранней стадии развития (т.е. незрелые В-клетки), зрелые В-клетки в процессе терминальной дифференцировки в плазматические клетки и злокачественные В-клетки. CD19 экспрессируется большинством пре-В острых лимфобластных лейкозов (ОЛЛ), неходжкинских лимфом, В-клеточных хронических лимфоцитарных лейкозов (ХЛЛ), пролимфоцитарных лейкозов, волосатоклеточных лейкозов, обычных острых лимфоцитарных лейкозов и некоторыми нуль-острых лимфобластных лейкозов. Экспрессия CD19 на плазматических клетках дополнительно позволяет предположить, что он может экспрессироваться на дифференцированных В-клеточных опухолях, таких как множественная миелома. Следовательно, антиген CD19 является мишенью для иммунотерапии при лечении неходжкинской лимфомы, хронического лимфоцитарного лейкоза и/или острого лимфобластного лейкоза.
«CD79b» относится к бета-цепи белка, ассоциированного с комплексом антигенного рецептора В-клеток, также известного как Ig-бета или специфический к В-клеткам гликопротеин В29, и включает любой нативный CD79b из любого источника; относящегося к позвоночным, включая млекопитающих, таких как приматы (например, люди), отличные от человека приматы (например, яванские макаки) и грызуны (например, мыши и крысы), если не указано иное. Аминокислотная последовательность CD79b человека приведена под номером доступа UniProt №Р40259 (версия 180, SEQ ID NO:435). CD79b представляет собой 39 кДа белок, экспрессируемый исключительно на В-клетках, и вместе с CD79a инициирует каскад сигнальной трансдукции после BCR, что приводит к интернализации комплекса BCR, его транслокации в эндосомы и презентации антигена. CD79 (состоящий из субъединиц CD79a и CD79b) представляет собой гетеродимерный компонент трансдукции сигналов В-клеточного рецептора, повсеместно экспрессируемый в зрелых В-клеточных лимфомах и размещаемый на поверхности клеток наиболее ранними предшественниками коммитированных В-клеток до экспрессии иммуноглобулина и. Термин «CD79b» охватывает «полноразмерный» непроцессированный CD79b, а также любую форму CD79b, полученную в результате процессинга в клетке. Этот термин также охватывает варианты CD79b природного происхождения, например, сплайс-варианты или аллельные варианты.
Термин «CD20» относится к В-лимфоцитарному антигену CD20, также известному как В-лимфоцитарный поверхностный антиген В1 или лимфоцитарный поверхностный антиген Leu-16, и включает любой нативный CD20 из любого источника, относящегося к позвоночным, включая млекопитающих, таких как приматы (например, люди), отличные от человека приматы (например, яванские макаки) и грызуны (например, мыши и крысы), если не указано иное. Аминокислотная последовательность CD20 человека приведена под номером доступа UniProt №Р11836 (версия 149, SEQ ID NO:436). CD20 представляет собой гидрофобный трансмембранный белок с молекулярной массой приблизительно 35 кДа, экспрессируемый на пре-В и зрелых В-лимфоцитах. Соответствующим человеческим геном является трансмембранный 4-доменный член 1 подсемейства А, также известный как MS4A1. Это ген кодирует члена семейства трансмембранных генов 4А. Члены этого растущего семейства белков характеризуются общими структурными признаками и схожими границами сплайсинга интрон/экзон и демонстрируют уникальные профили экспрессии среди гемопоэтических клеток и нелимфоидных тканей. Этот ген кодирует поверхностную молекулу В-лимфоцитов, которая играет роль в развитии и дифференцировке В-клеток в плазматические клетки. Это член локализован на 11q12, среди кластера членов семейства. Альтернативный сплайсинг этого гена приводит к двум вариантам транскрипта, которые кодируют один белок. Термин «CD20» охватывает «полноразмерный» непроцессированный CD20, а также любую форму CD20, полученную в результате процессинга в клетке. Этот термин также охватывает варианты CD20 природного происхождения, например, сплайс-варианты или аллельные варианты.
Термин «CD22» относится к В-клеточному рецептору CD22, также известному как В-лимфоцитарная молекула адгезии или SIGLEC2, и включает любой нативный CD22 из любого источника, относящегося к позвоночным, включая млекопитающих, таких как приматы (например, люди), отличные от человека приматы (например, яванские макаки) и грызуны (например, мыши и крысы), если не указано иное. Аминокислотная последовательность CD22 человека приведена под номером доступа UniProt №Р20273 (версия 209, SEQ ID NO:437). CD22 представляет собой молекулу, принадлежащую к семейству лектинов SIGLEC, и встречается на поверхности зрелых В-клеток и, в меньшей степени, на некоторых незрелых В-клетках. Таким образом, CD22 представляет собой ограниченный В-клетками фосфогликопротеин клеточной поверхности массой 130 150 кДа и способен модулировать опосредованные В-лимфоцитарным антигенным рецептором (BCR) сигналы, а также генерацию BCR-независимых сигналов. Термин «CD22» охватывает «полноразмерный» непроцессированный CD22, а также любую форму CD22, полученную в результате процессинга в клетке. Этот термин также охватывает варианты CD22 природного происхождения, например, сплайс-варианты или аллельные варианты.
«CD37» относится к лейкоцитарному антигену CD37, также известному как тетраспанин-26 (Tspan-26), и включает любой нативный CD37 из любого источника, относящегося к позвоночным, включая млекопитающих, таких как приматы (например, люди), отличные от человека приматы (например, яванские макаки) и грызуны (например, мыши и крысы), если не указано иное. Аминокислотная последовательность CD37 человека приведена под номером доступа UniProt №PI 1049 (версия 162, SEQ ID NO:438). Экспрессия CD37 ограничена клетками иммунной системы с наибольшей распространенностью на зрелых В-клетках и более низкой экспрессией на Т-клетках и миелоидных клетках. Гликопротеин CD37 является членом суперсемейства трансмембранных белков 4 и регулирует как гуморальные, так и клеточные иммунные ответы. Термин «CD37» охватывает «полноразмерный» непроцессированный CD37, а также любую форму CD37, полученную в результате процессинга в клетке. Этот термин также охватывает варианты CD37 природного происхождения, например, сплайс-варианты или аллельные варианты.
В контексте данного документа «антиген клеточной поверхности множественной миеломы (ММ)» относится к антигенной детерминанте, представленной на поверхности клеток множественной миеломы (ММ). Несколько антигенов клеточной поверхности ММ интересны в терминах иммунотерапии множественной миеломы. В одном аспекте антиген клеточной поверхности ММ выбран из группы, состоящей из CD38, ВСМА и GPRC5D.
Термин «CD38», также известный как кластер дифференцировки 38 или циклическая АДФ-рибоза гидролаза, представляет собой гликопротеин, встречающийся на поверхности многих иммунных клеток (белых кровяных клеток), включая CD4+, CD8+, В-лимфоциты и естественные клетки-киллеры. CD38 также функционирует в клеточной адгезии, сигнальной трансдукции и кальциевой сигнализации. В нормальных условиях CD38 экспрессируется на относительно низких уровнях на миелоидных и лимфоидных клетках и в некоторых не гемопоэтических тканях. В противоположность этому, нормальные плазматические клетки и клетки множественной миеломы (ММ) имеют высокие уровни экспрессии CD38, что делает CD38 интересной мишенью для нацеливания на молекулы клеточной поверхности при ММ. В контексте данного документа CD38 относится к любому белку CD38 из любого источника, относящегося к позвоночным, включая млекопитающих, таких как приматы (например, люди), отличные от человека приматы (например, яванские макаки) и грызуны (например, мыши и крысы), если не указано иное. Аминокислотная последовательность CD38 человека приведена под номером доступа UniProt (www.uniprot.org) №Р28907 (SEQ ID NO:474).
Термин «ВСМА» относится к антигену созревания В-клеток, также называемому членом 17 суперсемейства рецепторов фактора некроза опухоли (TNFRS17), и представляет собой трансмембранный белок III типа без сигнального пептида и содержит богатые цистеином внеклеточные домены. ВСМА экспрессируется на значительно больших уровнях во всех клетках ММ пациентов, но не в других нормальных тканях за исключением нормальных плазматических клеток. ВСМА, наряду с двумя родственными рецепторами, рецептором фактора активации В-клеток суперсемейства TNFR (BAFF-R) и трансмембранным активатором и партнером кальциевого модулятора и лиганда циклофилина (TACI), критически регулируют пролиферацию и выживаемость В-клеток, а также созревание и дифференцировку в плазматические клетки. Эти три функционально родственных рецептора поддерживают долгосрочную выживаемость В-клеток на разных стадиях развития путем связывания с BAFF и/или APRIL, их когнатными лигандами. В контексте данного документа ВСМА относится к любому белку ВСМА из любого источника, относящегося к позвоночным, включая млекопитающих, таких как приматы (например, люди), отличные от человека приматы (например, яванские макаки) и грызуны (например, мыши и крысы), если не указано иное. Аминокислотная последовательность ВСМА человека приведена под номером доступа UniProt (www.uniprot.org) №Q02223 (SEQ ID NO:475).
Термин «GPRC5D» относится к сопряженному с G-белком рецептору, класс С, группа 5, член D, мишени, идентифицированной из плазматических клеток в множественной миеломе с помощью РНК-секвенирования. Сообщалось, что GPRC5D связан с плохим прогнозом и опухолевой нагрузкой у пациентов с множественной миеломой. GPRC5D относится к любому белку GPRC5D из любого источника, относящегося к позвоночным, включая млекопитающих, таких как приматы (например, люди), отличные от человека приматы (например, яванские макаки) и грызуны (например, мыши и крысы), если не указано иное. Аминокислотная последовательность GPRC5D человека приведена под номером доступа UniProt (www.uniprot.org) №Q9NZD1 (SEQ ID NO:476).
Термин «CD28» (кластер дифференцировки 28, Тр44) относится к любому белку CD28 из любого источника, относящегося к позвоночным, включая млекопитающих, таких как приматы (например, люди), отличные от человека приматы (например, яванские макаки) и грызуны (например, мыши и крысы), если не указано иное. CD28 экспрессируется на Т-клетках и обеспечивает костимулирующие сигналы, необходимые для активации и выживаемости Т-клеток. Стимуляция Т-клеток посредством CD28 в дополнение к Т-клеточному рецептору (ТКР) может обеспечить активный сигнал для выработки различных интерлейкинов. CD28 является рецептором для белков CD80 (В7.1) и CD86 (В7.2) и единственным рецептором В7, который конститутивно экспрессируется на наивных Т-клетках. Аминокислотная последовательность CD28 человека приведена под номером доступа UniProt (www.uniprot.org) №Р 10747 (SEQ ID NO:1).
«Агонистическое антитело» относится к антителу, которое имеет агонистическую функцию в отношении заданного рецептора. В общем случае, когда агонистический лиганд (фактор) связывается с рецептором, меняется третичная структура рецепторного белка и происходит активация рецептора (когда рецептор представляет собой мембранный белок, обычно происходит трансдукция сигнала клеточного роста или подобного сигнала). Если рецептор принадлежит к димер-образующему типу, агонистическое антитело может димеризовать рецептор с соответствующими расстоянием и углом, действуя, таким образом, аналогично лиганду. Соответствующее антитело против рецептора может имитировать димеризацию рецепторов, осуществляемую лигандами, и, таким образом, становится агонистическим антителом.
«Агонистическая CD28-антигенсвязывающая молекула» или «стандартная агонистическая CD28-антигенсвязывающая молекула» представляет собой антигенсвязывающую молекулу, которая имитирует природные лиганды CD28 (CD80 или CD86) в отношении их роли в усилении активации Т-клеток в присутствии сигнала Т-клеточного рецептора («сигнал 2»). Для полной активации Т-клеток необходимы два сигнала. В физиологических условиях «сигнал 1» возникает в результате взаимодействия молекул Т-клеточного рецептора (ТКР) с комплексами пептид/главный комплекс гистосовместимости (ГКС) на антигенпрезентирующих клетках (АПК), а «сигнал 2» обеспечивает привлечение костимулирующего рецептора, например, CD28. Агонистическая CD28-антигенсвязывающая молекула способна костимулировать Т-клетки (сигнал 2). Также она способна индуцировать пролиферацию Т-клеток и секрецию цитокинов в комбинации с молекулой со специфичностью в отношении комплекса ТКР, однако агонистическая CD28-антигенсвязывающая молекула не способна полностью активировать Т-клетки без дополнительной стимуляции ТКР. При этом существует подкласс агонистических CD28-антигенсвязывающих молекул, так называемые суперагонистические CD28-антигенсвязывающие молекулы. «Суперагонистическая CD28-антигенсвязывающая молекула» представляет собой CD28-антигенсвязывающую молекулу, которая способна полностью активировать Т-клетки без дополнительной стимуляции ТКР. Суперагонистическая CD28-антигенсвязывающая молекула способна индуцировать пролиферацию Т-клеток и секрецию цитокинов без предварительной активации Т-клеток (сигнал 1).
Термин «вариабельный домен» или «вариабельная область» относится к домену тяжелой или легкой цепи антитела, который участвует в связывании антигенсвязывающей молекулы с антигеном. Вариабельные домены тяжелой цепи и легкой цепи (VH и VL, соответственно) нативного антитела обычно имеют сходные структуры, причем каждый домен содержит четыре консервативные каркасные области (FR) и три гипервариабельные области (HVR). Смотрите, например, Kindt et al., Kuby Immunology, 6th ed., W.H. Freeman and Co., страница 91 (2007). Одного домена VH или VL может быть достаточно для придания антигенсвязывающей специфичности.
В контексте данного документа термин «гипервариабельная область» или «HVR» относится к каждой из областей антигенсвязывающего вариабельного домена, которые являются гипервариабельными по последовательности и которые определяют антигенсвязывающую специфичность, например, как «определяющие комплементарность области» («CDR»). В общем случае антигенсвязывающие домены содержат шесть CDR: три в VH (CDR-H1, CDR-H2, CDR-H3) и три в VL (CDR-L1, CDR-L2, CDR-L3). Типовые CDR по данному документу включают:
(а) гипервариабельные петли, находящиеся в аминокислотных остатках 26-32 (L1), 50-52 (L2), 91-96 (L3), 26-32 (H1), 53-55 (H2) и 96-101 (Н3) (Chothia and Lesk, J. Mol. Biol. 196:901-917 (1987));
(б) CDR, находящиеся в аминокислотных остатках 24-34 (L1), 50-56 (L2), 89-97 (L3), 31-35b (H1), 50-65 (H2) и 95-102 (Н3) (Kabat et al.. Sequences of Proteins of Immunological Interest, 5th Ed. Public Health Service, National Institutes of Health, Bethesda, MD (1991)); и
(в) антигенные контакты, находящиеся в аминокислотных остатках 27 с 36 (L1), 46-55 (L2), 89-96 (L3), 30-35b (H1), 47-58 (H2) и 93-101 (Н3) (MacCallum et al. J. Mol. Biol. 262: 732-745 (1996)).
Если не указано иное, CDR определены в соответствии с Kabat et al., выше. Специалисту в данной области техники понятно, что обозначения CDR также могут быть определены в соответствии с Chothia, выше, McCallum, выше, или любой другой научно приемлемой номенклатурой. В Kabat et al. также приведено определение системы нумерации для последовательностей вариабельной области, которая применима к любому антителу. Специалист в данной области техники может однозначно применить эту систему «нумерации по Kabat» к любой последовательности вариабельной области независимо от каких-либо экспериментальных данных помимо самой последовательности. В контексте данного документа «нумерация по последовательностей вариабельной области» относится к системе нумерации, установленной в Kabat et al., U.S. Dept. of Health and Human Services, "Sequence of Proteins of Immunological Interest" (1983). Если не указано иное, ссылки на нумерацию позиций конкретных аминокислотных остатков в вариабельной области антитела сделаны в соответствии с системой нумерации по Kabat.
В данном документе термин «с созревшей аффинностью» в контексте антигенсвязывающих молекул (например, антител) относится к антигенсвязывающей молекуле, которая получена из референсной антигенсвязывающей молекулы, например, посредством мутации, связывается с тем же антигеном, предпочтительно связывается с тем же эпитопом, что и референсное антитело; и имеет большую аффинность в отношении антигена, чем референсная антигенсвязывающая молекула. Созревание аффинности в общем случае включает модификацию одного или более аминокислотных остатков в одной или более CDR антигенсвязывающей молекулы. Так правило, антигенсвязывающая молекула с созревшей аффинностью связывается с тем же эпитопом, что и исходная референсная антигенсвязывающая молекула.
«Каркасная область» или «FR» относится к остаткам вариабельного домена, отличным от остатков гипервариабельной области (HVR). FR вариабельного домена обычно состоит из четырех FR-доменов: FR1, FR2, FR3 и FR4. Соответственно, последовательности HVR и FR обычно находятся в VH (или VL) в следующем порядке: FR1-H1(L1)-FR2-H2(L2)-FR3-H3(L3)-FR4.
В целях данного документа «человеческая акцепторная каркасная область» представляет собой каркасную область, содержащую аминокислотную последовательность каркасной области вариабельного домена легкой цепи (VL) или каркасной области вариабельного домена тяжелой цепи (VH), полученную из каркасной области человеческого иммуноглобулина или человеческой консенсусной каркасной области, как определено ниже. Человеческая акцепторная каркасная область, «полученная из» каркасной области человеческого иммуноглобулина или человеческой консенсусной каркасной области, может содержать такую же самую аминокислотную последовательность или может содержать изменения в аминокислотной последовательности. В некоторых вариантах осуществления число аминокислотных изменений составляет 10 или менее, 9 или менее, 8 или менее, 7 или менее, 6 или менее, 5 или менее, 4 или менее, 3 или менее, или 2 или менее. В некоторых вариантах осуществления человеческая акцепторная каркасная область VL идентична по последовательности с последовательностью каркасной области VL человеческого иммуноглобулина или последовательностью человеческой консенсусной каркасной области.
Термин «химерное» антитело относится к антителу, в котором часть тяжелой и/или легкой цепи получена из определенного источника или вида, тогда как остальная часть тяжелой и/или легкой цепи получена из другого источника или вида.
«Класс» антитела относится к типу константного домена или константной области, которые содержит его тяжелая цепь. Существует пять основных классов антител: IgA, IgD, IgE, IgG и IgM, а некоторые из них можно дополнительно разделить на подклассы (изотипы), например, IgG1, IgG2, IgG3, IgG4, IgA1 и IgA2. Константные домены тяжелой цепи, соответствующие разным классам иммуноглобулинов, называются α, δ, ε, γ и μ, соответственно.
«Гуманизированное» антитело относится к химерному антителу, содержащему аминокислотные остатки из нечеловеческих HVR и аминокислотные остатки из человеческих FR. В определенных вариантах осуществления гуманизированное антитело содержит практически все из по меньшей мере одного и, как правило, двух вариабельных доменов, в которых все или практически все HVR (например, CDR) соответствуют таковым из нечеловеческого антитела, а все или практически все FR соответствуют таковым из человеческого антитела. Гуманизированное антитело, необязательно, может содержать по меньшей мере часть константной области антитела, полученную из человеческого антитела. «Гуманизированная форма» антитела, например, нечеловеческого антитела, относится к антителу, подвергшемуся гуманизации. Другие формы «гуманизированных антител», охватываемые настоящим изобретением, представляют собой те, в которых константная область была дополнительно модифицирована или изменена относительно исходного антитела для обеспечения свойств в соответствии с изобретением, в особенности в отношении связывания C1q и/или связывания Fc-рецептора (FcR).
«Человеческое» антитело представляет собой антитело, которое имеет аминокислотную последовательность, соответствующую последовательности антитела, вырабатываемого человеком или клеткой человека, или полученную из источника, отличного от человека, в котором используются репертуары человеческих антител или другие последовательности, кодирующие человеческие антитела. Это определение человеческого антитела явным образом исключает гуманизированное антитело, содержащее нечеловеческие антигенсвязывающие остатки.
Термин «домен СН1» обозначает часть полипептида тяжелой цепи антитела, которая простирается приблизительно от позиции 118 по EU до позиции 215 по EU (система нумерации EU в соответствии с Kabat). В одном аспекте домен СН1 имеет аминокислотную последовательность ASTKGPSVFP LAPSSKSTSG GTAALGCLVK DYFPEPVTVS WNSGALTSGV HTFPAVLQSS GLYSLSSVVT VPSSSLGTQT YICNVNHKPS NTKVDKKV (SEQ ID NO: 477). Обычно за ней следует сегмент, имеющий аминокислотную последовательность EPKSC (SEQ ID NO: 480), связывающий домен СН1 с шарнирной областью.
Термин «шарнирная область» обозначает часть полипептида тяжелой цепи антитела, которая в тяжелой цепи антитела дикого типа соединяет домен СН1 и домен СН2, например, приблизительно от позиции 216 приблизительно до позиции 230 в соответствии с системой нумерации EU по Kabat, или приблизительно от позиции 226 приблизительно до позиции 230 в соответствии с системой нумерации EU по Kabat. Шарнирные области других подклассов IgG можно определить путем выравнивания с остатками цистеина шарнирной области последовательности подкласса IgG1. Шарнирная область обычно представляет собой димерную молекулу, состоящую из двух полипептидов с идентичной аминокислотной последовательностью. В общем случае шарнирная область содержит до 25 аминокислотных остатков и является гибкой, позволяя связанным с ней связывающим мишень сайтам двигаться независимо. Шарнирную область можно разделить на три домена: верхний, средний и нижний шарнирные домены (смотрите, например, Roux, et al., J. Immuno1. 161 (1998) 4083).
В одном аспекте шарнирная область имеет аминокислотную последовательность DKTHTCPXCP (SEQ ID NO: 481), где X представляет собой S или Р. В одном аспекте шарнирная область имеет аминокислотную последовательность НТСРХСР (SEQ ID NO: 482), где X представляет собой S или Р. В одном аспекте шарнирная область имеет аминокислотную последовательность СРХСР (SEQ ID NO: 483), где Х представляет собой S или Р.
В данном документе термин «Fc-домен» или «Ес-область» используется для определения С-концевой области тяжелой цепи антитела, которая содержит по меньшей мере часть константной области. Этот термин включает Ес-области с нативной последовательностью и вариантные Ес-области. Ес-область IgG содержит СН2-домен IgG и СН3-домен IgG.
«СН2-домен» Ес-области IgG человека обычно простирается от аминокислотного остатка приблизительно в позиции 231 по EU до аминокислотного остатка приблизительно в позиции 340 по EU (система нумерации EU no Kabat). В одном аспекте домен СН2 имеет аминокислотную последовательность APELLGGPSV FLFPPKPKDT LMISRTPEVT CVWDVSHEDP EVKFNWYVDG VEVHNAKTKP REEQESTYRW SVLTVLHQDW LNGKEYKCKV SNKALPAPIE KTISKAK (SEQ ID NO: 478). Домен СН2 является уникальным в том, что он не характеризуется близким спариванием с другим доменом. Точнее, две N-связанные разветвленные углеводные цепи расположены между двумя доменами СН2 интактной нативной Ес-области. Было предположено, что этот углевод может заменять домен-доменное спаривание и помогать стабилизировать домен СН2. Burton, Mol. Immunol. 22 (1985) 161-206. В одном варианте осуществления к СН2-домену присоединена углеводная цепь. В данном документе СН2-домен может представлять собой нативный СН2-домен или вариантный СН2-домен.
Термин «домен СН3» содержит участок из остатков, расположенных С-терминально к домену СН2 в Ес-области и обозначает часть полипептида тяжелой цепи антитела, которая простирается приблизительно от позиции 341 по EU до позиции 446 по EU (система нумерации EU в соответствии с Kabat). В одном аспекте домен СН3 имеет аминокислотную последовательность GQPREPQVYT LPPSRDELTK NQVSLTCLVK GFYPSDIAVE WESNGQPENN YKTTPPVLDS DGSFFLYSKL TVDKSRWQQG NVFSCSVMHE ALHNHYTQKS LSLSPG (SEQ ID NO: 479). В данном документе СН3-область может представлять собой СН3-домен с нативной последовательностью или вариантный СН3-домен (например, СН3-домен с внесенным в одну цепь «выступом» («узлом») и соответствующей внесенной в другую цепь «полостью» («впадиной»); смотрите патент США 5821333, явным образом включенный в данный документ посредством ссылки). Такие вариантные СН3-домены можно использовать, чтобы стимулировать гетеродимеризацию двух неидентичных тяжелых цепей антитела, как описано в данном документе. В одном варианте осуществления Fc-область тяжелой цепи человеческого IgG простирается от Cys226 или от Pro230 до карбокси-конца тяжелой цепи. При этом С-концевой лизин (Lys447) Fc-области может присутствовать или нет. Если в данном документе не указано иное, нумерация аминокислотных остатков в Fc-области или константной области соответствует системе нумерации EU, также называемой индексом EU, как описано в Kabat et al.. Sequences of Proteins of Immunological Interest, 5th Ed. Public Health Service, National Institutes of Health, Bethesda, MD, 1991.
Технология «выступ-во-впадину» описана, например, в US 5731168; US 7695936; Ridgway et al., Prot Eng 9, 617-621 (1996) и Carter, J Immunol Meth 248, 7-15 (2001). В общем случае этот способ включает внесение выступа («узла») в контактную поверхность первого полипептида и соответствующей впадины («полости») в контактную поверхность второго полипептида, так что выступ может располагаться во впадине так, чтобы стимулировать образование гетеродимера и затруднять образование гомодимера. Выступы конструируют путем замены небольших аминокислотных боковых цепей в контактной поверхности первого полипептида на более крупные боковые цепи (например тирозин или триптофан). Компенсаторные «полости» идентичного или сходного размера с выступами создают в контактной поверхности второго полипептида путем замещения крупных аминокислотных боковых цепей меньшими (например, аланином или треонином). Выступ и полость можно создавать путем изменения нуклеиновой кислоты, кодирующей полипептиды, например, с помощью сайт-специфического мутагенеза, или с помощью пептидного синтеза. В конкретном варианте осуществления модификация узла включает аминокислотную замену T366W в одной из двух субъединиц Fc-домена, а модификация впадины включает аминокислотные замены T366S, L368A и Y407V в другой из двух субъединиц Fc-домена. В дополнительном конкретном варианте осуществления субъединица Fc-домена, содержащая модификацию выступа, дополнительно содержит аминокислотную замену S354C, а субъединица Fc-домена, содержащая модификацию впадины, дополнительно содержит аминокислотную замену Y349C. Внесение этих двух остатков цистеина приводит к образованию дисульфидного мостика между двумя субъединицами Fc-области, таким образом, дополнительно стабилизируя димер (Carter, J Immunol Methods 248, 7-15 (2001)).
Предполагается, что «область, эквивалентная Fc-области иммуноглобулина» включает встречающиеся в природе аллельные варианты Ес-области иммуноглобулина, а также варианты, имеющие изменения, которые соответствуют заменам, добавлениям или делециям, но которые практически не снижают способность иммуноглобулина опосредовать эффекторные функции (такие как антителозависимая клеточная цитотоксичность). Например, из N-конца или С-конца Fc-области иммуноглобулина могут быть удалены одна или более аминокислот практически без утраты биологической функции. Такие варианты можно отбирать в соответствии с общими правилами, известными в данной области техники, как имеющие минимальный эффект на активность (смотрите, например, Bowie, J. U. et al., Science 247:1306-10 (1990)).
«Fc-домен дикого типа» обозначает аминокислотную последовательность, идентичную аминокислотной последовательности Fc-домена, встречающейся в природе. Fc-домены дикого типа включают нативную Fc-область IgG1 человека (аллотипы не-А и А); нативную Fc-область IgG2 человека; нативную Fc-область IgG3 человека; и нативную Fc-область IgG4 человека, а также их встречающиеся в природе варианты. Fc-области дикого типа обозначены в SEQ ID NO: 484 (IgG1, европеоидный аллотип), SEQ ID NO: 485 (IgG1, афроамериканский аллотип), SEQ ID NO: 486 (IgG2), SEQ ID NO: 487 (IgG3) и SEQ ID NO: 488 (IgG4).
Термин «вариантный Fc-домен (человека)» обозначает аминокислотную последовательность, которая отличается от аминокислотной последовательности Fc-домена (человека) «дикого типа» за счет по меньшей мере одной «аминокислотной мутации». В одном аспекте вариантная Fc-область имеет по меньшей мере одну аминокислотную мутацию по сравнению с нативной Fc-областью, например, от около одной до около десяти аминокислотных мутаций, а в одном аспекте от около одной до около пяти аминокислотных мутаций в нативной Fc-области. В одном аспекте (вариантная) Fc-область имеет по меньшей мере около 95% гомологии с Fc-областью дикого типа.
Термин «эффекторные функции» относится видам биологической активности, характерным для Fc-области антитела, которые варьируются в зависимости от изотипа антитела. Примеры эффекторных функций антител включают: связывание C1q и комплементзависимую цитотоксичность (КЗЦ), связывание Fc-рецептора, антителозависимую клеточноопосредованную цитотоксичность (АЗКЦ), антителозависимый клеточный фагоцитоз (АЗКФ), секрецию цитокинов, опосредованное иммунных комплексом поглощение антигена антигенпрезентирующими клетками, понижающую регуляцию рецепторов клеточной поверхности (например, В-клеточного рецептора) и активацию В-клеток.
Эффекторные функции связывания Fc-рецептора могут быть опосредованы взаимодействием Fc-области антитела с Fc-рецепторами (FcR), которые представляют собой специализированный рецепторы клеточной поверхности на гемопоэтических клетках. Fc-рецепторы принадлежат к суперсемейству иммуноглобулинов, и было показано, что они опосредуют как удаление покрытых антителами патогенов посредством фагоцитоза иммунных комплексов, так и лизис эритроцитов и различных других клеточных мишеней (например, опухолевых клеток), покрытых соответствующими антителами, посредством антителозависимой клеточноопосредованной цитотоксичности (АЗКЦ) (смотрите, например. Van de Winkel, J.G. and Anderson, C.L., J. Leukoc. Biol. 49 (1991) 511-524). FcR определяют по их специфичности в отношении изотипов иммуноглобулина: Fc-рецепторы для антител IgG называются FcγR. Связывание Fc-рецепторов описано, например, в Ravetch, J.V. and Kinet, J.P., Annu. Rev. Immunol. 9 (1991) 457-492; Capel, P.J., et al., Immunomethods 4 (1994) 25-34; de Haas, M., et al., J. Lab. Clin. Med. 126 (1995) 330-341; и Gessner, J.E., et al., Ann. Hematol. 76 (1998) 231-248.
Перекрестное связывание рецепторов к Fc-области антител IgG (FcγR) стимулирует широкий ряд эффекторных функций, включая фагоцитоз, антителозависимую клеточную цитотоксичность и высвобождение воспалительных медиаторов, а также выведение иммунных комплексов и регуляцию выработки антител. У людей были описаны три класса FcγR, которые представляют собой:
- FcγRI (CD64) связывает мономерный IgG с высокой аффинностью и экспрессируется на макрофагах, моноцитах, нейтрофилах и эозинофилах. Модификация Fc-области IgG по меньшей мере в одном из аминокислотных остатков E233-G236, Р238, D265, N297, А327 и Р329 (нумерация в соответствии с индексом EU по Kabat) снижает связывание с FcγRI. Остатки IgG2 в позициях 233-236, замещенные на IgG1 и IgG4, снижали связывание с FcγRI в 103 раз и устраняли ответ моноцитов человека на сенсибилизированные антителом красные кровяные клетки (Armour, K.L., et al., Fur. J. Immunol. 29 (1999) 2613 2624).
- FcγRII (CD32) связывает комплексированный IgG со средней или низкой аффинностью и экспрессируется повсеместно. Этот рецептор можно разделить на два подтипа - FcγRIIA и FcγRIIB. FcγRIIA встречается на многих клетках, участвующих в уничтожении (например, макрофагах, моноцитах, нейтрофилах) и, похоже, способен активировать процесс уничтожения. FcγRIIB, похоже, играет роль в процессе ингибирования и встречается на В-клетках, макрофагах и на тучных клетках и эозинофилах. В случае В-клеток его функция, по-видимому, состоит в подавлении дальнейшей выработки иммуноглобулина и переключении изотипа, например, на класс IgE. В случае макрофагов действие FcγRIIB состоит в ингибировании фагоцитоза, опосредованного FcγRIIA. В случае эозинофилов и тучных клеток В-форма может способствовать подавлению активации этих клеток посредством связывания IgE с отдельным рецептором. Сниженное связывание с FcγRIIA встречается, например, у антител, содержащих Fc-область IgG с мутациями по меньшей мере в одном из аминокислотных остатков E233-G236, Р238, D265, N297, А327, Р329, D270, Q295, А327, R292 и K414 (нумерация в соответствии с индексом EU по Kabat).
- FcγRIII (CD16) связывает IgG со средней или низкой аффинностью и существует в виде двух типов. FcγRIIIA встречается на NK-клетках, макрофагах, эозинофилах и некоторых моноцитах и Т-клетках и опосредует АЗКЦ. FcγRIIIB на высоком уровне экспрессируется на нейтрофилах. Сниженное связывание с FcγRIIIA встречается, например, у антител, содержащих Fc-область IgG с мутациями по меньшей мере в одном из аминокислотных остатков E233-G236, Р238, D265, N297, А327, Р329, D270, Q295, А327, S239, Е269, Е293, Y296, V303, А327, K338 и D376 (нумерация в соответствии с индексом EU по Kabat).
Картирование сайтов связывания с Fc-рецепторами на человеческом IgG1, вышеуказанные сайты мутации и способы измерения связывания с FcγRI и FcγRIIA описаны в Shields, R.L., et al. J. Biol. Chem. 276 (2001) 6591-6604.
Термин «АЗКЦ» или «антителозависимая клеточная цитотоксичность» обозначает иммунный механизм, приводящий к лизису покрытых антителами клеток-мишеней иммунными эффекторными клетками. Клетки-мишени (целевые клетки) представляют собой клетки, с которыми специфически связываются антитела или их производные, содержащие Fc-область, обычно посредством белковой части, расположенной N-терминально к Fc-области. В контексте данного документа термин «сниженная АЗКЦ» определяется как снижение числа клеток-мишеней, лизируемых за заданное время при заданной концентрации антитела в среде, окружающей клетки-мишени, за счет механизма АЗКЦ, определенного выше, и/или как повышение концентрации антитела в среде, окружающей клетки-мишени, необходимой для обеспечения лизиса заданного числа клеток-мишеней за заданное время за счет механизма АЗКЦ. Снижение АЗКЦ определяют относительно АЗКЦ, опосредованной тем же антителом, вырабатываемым таким же типом клеток-хозяев, с использованием таких же стандартных методов получения, очистки, составления и хранения (которые известны специалистам в данной области техники), но которое не было сконструировано. Например, снижение АЗКЦ, опосредованной антителом, содержащим в Fc-домене аминокислотную замену, которая снижает АЗКЦ, определяют относительно АЗКЦ, опосредованной таким же антителом без этой аминокислотной замены в Fc-домене. Подходящие анализы для измерения АЗКЦ хорошо известны в данной области техники (смотрите, например, публикацию РСТ № WO 2006/082515 или публикацию РСТ № WO 2012/130831). Например, способность антител индуцировать начальные этапы опосредования АЗКЦ исследуют путем измерения их связывания с экспрессирующими Fcγ-рецепторы клетками, такими как клетки, рекомбинантно экспрессирующие FcγRI и/или FcγRIIA, или NK-клетки (экспрессирующие преимущественно FcγRIIIA). В частности, измеряют связывание с FcγR на NK-клетках.
«Активирующий Fc-рецептор» представляет собой Ес-рецептор, который после связывания Fc-областью антитела вызывает сигнальные события, которые стимулируют несущие рецепторы клетки осуществлять эффекторные функции. Активирующие Fc-рецепторы включают FcγRIIIa (CD16a), FcγRI (CD64), FcγRIIa (CD32) и FcαRI (CD89). Конкретным активирующим Fc-рецептором является человеческий FcγRIIIa (смотрите номер доступа UniProt Р08637, версия 141).
«Эктодомен» представляет собой мембранный белок, который простирается во внеклеточное пространство (т.е. пространство за пределами клетки). Эктодомены обычно являются частями белков, которые инициируют контакт с поверхностями, что приводит к трансдукции сигнала.
Термин «пептидный линкер» относится к пептиду, содержащему одну или более аминокислот, как правило, приблизительно 2-20 аминокислот. Пептидные линкеры известны в данной области техники или описаны в данном документе. Подходящими неиммуногенными линкерными пептидами являются, например, пептидные линкеры (G4S)n, (SG4)n или G4(SG4)n, где «n» в общем случае представляет собой число от 1 и 5, как правило, от 2 до 4, в частности 2, т.е. пептиды, выбранные из группы, состоящей из GGGGS (SEQ ID NO: 146), GGGGSGGGGS (SEQ ID NO: 147), SGGGGSGGGG (SEQ ID NO: 148) и GGGGSGGGGSGGGG (SEQ ID NO: 149), но также включают последовательности GSPGSSSSGS (SEQ ID NO: 150), (G4S)3 (SEQ ID NO: 151), (G4S)4 (SEQ ID NO: 152), GSGSGSGS (SEQ ID NO: 153), GSGSGNGS (SEQ ID NO: 154), GGSGSGSG (SEQ ID NO: 155), GGSGSG (SEQ ID NO: 156), GGSG (SEQ ID NO: 157), GGSGNGSG (SEQ ID NO: 158), GGNGSGSG (SEQ ID NO: 159) и GGNGSG (SEQ ID NO: 160). Особенный интерес представляют пептидные линкеры (G4S) (SEQ ID NO: 146), (G4S)2 или GGGGSGGGGS (SEQ ID NO: 147), (G4S)3 (SEQ ID NO: 151) и (G4S)4 (SEQ ID NO: 152).
В контексте данной заявки термин «аминокислота» обозначает группу α-аминокислот природного происхождения, включая аланин (трехбуквенный код: ala, однобуквенный код: А), аргинин (arg, R), аспарагин (asn, N), аспарагиновая кислота (asp, D), цистеине (cys, С), глутамин (gln, Q), глутаминовая кислота (glu, Е), глицин (gly, G), гистидин (his, H), изолейцин (ile, I), лейцин (leu, L), лизин (lys, K), метионин (met, М), фенилаланин (phe, F), пролин (pro, Р), серии (ser, S), треонин (thr, Т), триптофан (trp, W), тирозин (tyr, Y) и валин (val, V).
Под «слитым» или «соединенным» подразумевается, что компоненты (например, полипептид или эктодомен указанного члена семейства лигандов TNF) связаны пептидными связями, как напрямую, так и посредством одного или более пептидных линкеров.
«Процент (%) идентичности аминокислотной последовательности» относительно референсной полипептидной (белковой) последовательности определяется как процентная доля аминокислотных остатков в кандидатной последовательности, которые являются идентичными с аминокислотными остатками в референсной полипептидной последовательности, после выравнивания последовательностей и внесения, в случае необходимости, гэпов для достижения максимального процента идентичности последовательностей, и без учета каких-либо консервативных замен в качестве части идентичности последовательностей. Выравнивание в целях определения процента идентичности аминокислотной последовательности можно осуществлять различными способами, которые известны в данной области техники, например, используя общедоступное компьютерное программное обеспечение, такое как программное обеспечение BLAST, BLAST-2, ALIGN, SAWI или Megalign (DNASTAR). Специалисты в данной области техники могут определить подходящие параметры для выравнивания последовательностей, включая любые алгоритмы, необходимые для достижения максимального выравнивания по всей длине сравниваемых последовательностей. При этом в целях данного документа значения процента (%) идентичности аминокислотных последовательностей получают, используя компьютерную программу для сравнения последовательностей ALIGN-2. Компьютерная программа для сравнения последовательностей ALIGN-2 была разработана Genentech, Inc., а исходный код был подан вместе с пользовательской документацией в Бюро регистрации авторских прав США, Вашингтон, округ Колумбия, 20559, где он зарегистрирован под номером регистрации авторского права США TXU510087. Программа ALIGN-2 находится в свободном доступе от Genentech, Inc., Южный Сан-Франциско, Калифорния, или может быть скомпилирована из исходного кода. Программу ALIGN-2 следует компилировать для использования в операционной системе UNIX, включая Digital UNIX V4.0D. Все параметры сравнения последовательностей установлены программой ALIGN-2 и не варьируются. В тех случаях, когда для сравнения аминокислотных последовательностей используют ALIGN-2, % идентичности аминокислотной последовательности заданной аминокислотной последовательности А с или по сравнению с заданной аминокислотной последовательностью В (что в альтернативном варианте можно сформулировать как заданная аминокислотная последовательность А, которая имеет или содержит определенный % идентичности аминокислотной последовательности с или по сравнению с заданной аминокислотной последовательностью В) рассчитывают следующим образом: 100 умножить на отношение X/Y, где Х представляет число аминокислотных остатков, оцененных программой выравнивания последовательностей ALIGN-2 как идентичные совпадения в осуществленном программой выравнивании А и В, a Y представляет общее число аминокислотных остатков в В. Следует понимать, что если длина аминокислотной последовательности А не равна длине аминокислотной последовательности В, % идентичности аминокислотной последовательности А с В не будет равен % идентичности аминокислотной последовательности В с А. Если специально не указано иное, все значения % идентичности аминокислотных последовательностей, используемые в данном документе, получены, как описано в предыдущем параграфе, с использованием компьютерной программы ALIGN-2.
В определенных вариантах осуществления предусмотрены варианты аминокислотной последовательности CD28-антигенсвязывающих молекул, предложенных в данном документе. Например, может существовать необходимость в улучшении аффинности связывания и/или других биологических свойств CD28-антигенсвязывающих молекул. Варианты аминокислотной последовательности CD28-антигенсвязывающих молекул можно получать путем внесения соответствующих модификаций в нуклеотидную последовательность, кодирующую молекулы, или с помощью пептидного синтеза. Такие модификации включают, например, делеции, и/или вставки, и/или замены остатков в аминокислотных последовательностях антитела. Для получения конечной конструкции можно осуществлять любую комбинацию делеции, вставок и замен при условии, что конечная конструкция обладает необходимыми характеристиками, например, связывания антигена.
Сайты, представляющие интерес для заместительного мутагенеза, включают HVR и каркасные области (FR). Консервативные замены приведены в таблице Б под заголовком «Предпочтительные замены» и дополнительно описаны ниже в отношении классов боковых цепей аминокислот от (1) до (6). Аминокислотные замены можно вносить в представляющую интерес молекулу и проводить скрининг продуктов в отношении необходимой активности, например, сохранения/улучшения связывания антигена, снижения иммуногенности или улучшения АЗКЦ или КЗЦ.
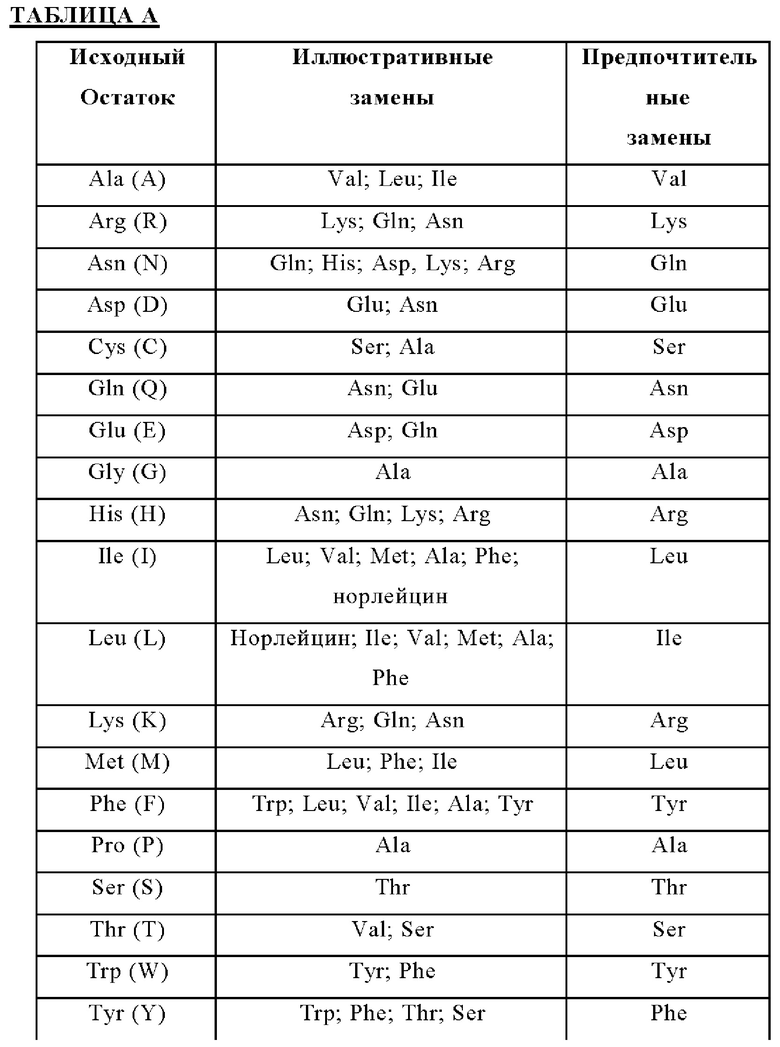

Аминокислоты можно разделить на группы в соответствии с общими свойствами боковых цепей:
(1) гидрофобные: норлейцин, Met, Ala, Val, Leu, Ile;
(2) нейтральные гидрофильные: Cys, Ser, Thr, Asn, Gln;
(3) кислые: Asp, Glu;
(4) основные: His, Lys, Arg;
(5) остатки, влияющие на ориентацию цепи: Gly, Pro;
(6) ароматические: Trp, Tyr, Phe.
Неконсервативные замены подразумевают замену представителя одного из этих классов представителем другого класса.
Термин «варианты аминокислотной последовательности» включает заместительные варианты, в которых аминокислотные замены присутствуют в одном или более остатках гиперварибельной области родительской антигенсвязывающей молекулы (например, гуманизированное или человеческое антитело). В общем случае полученные в результате варианты, отобранные для дополнительного исследования, имеют модификации (например, улучшение) определенных биологических свойств (например, повышенную аффинность, сниженную иммуногенность) по сравнению с родительской антигенсвязывающей молекулой и/или в значительной степени сохраняют определенные биологические свойства родительской антигенсвязывающей молекулы. Типовой заместительный вариант представляет собой антитело с созревшей аффинностью, которое удобно получать, например, используя методики созревания аффинности на основе фагового дисплея, такие как описанные в данном документе. Вкратце, один или более остатков HVR подвергают мутации, а вариантные антигенсвязывающие молекулы отображают на поверхности фага и проводят их скрининг в отношении конкретного вида биологической активности (например, аффинности связывания). В определенных вариантах осуществления замены, вставки или делеции могут находится в одной или более HVR при условии, что такие изменения практически не снижают способность антигенсвязывающей молекулы связывать антиген. Например, в HVR можно проводить консервативные изменения (например, консервативные замены, предложенные в данном документе), которые существенно не снижают аффинность связывания. Удобный способ идентификации остатков или областей антитела, которые могут быть мишенями для мутагенеза, называется «аланин-сканирующим мутагенезом», описанным Cunningham and Wells (1989) Science, 244:1081-1085. В этом способе идентифицируют аминокислотный остаток или группу целевых аминокислотных остатков (например, заряженные аминокислотные остатки, такие как Arg, Asp, His, Lys и Glu) и заменяют нейтральной или отрицательно заряженной аминокислотой (например, аланином или полиаланином) для определения, повлияло ли это на взаимодействие антитела с антигеном. Дополнительные замены можно вносить в аминокислотные положения, демонстрирующие функциональную чувствительность к исходным заменам. В альтернативном или дополнительном варианте используют кристаллическую структуру комплекса антигенсвязывающая молекула - антиген, чтобы идентифицировать точки контакта между антителом и антигеном. Такие контактные остатки и соседние остатки можно использовать для нацеливания или удалять из кандидатов для замены. Можно проводить скрининг вариантов для определения наличия у них необходимых свойств.
Вставки аминокислотных последовательностей включают амино- и/или карбокси-концевые слияния с диапазоном длины от одного остатка до полипептидов, содержащих сто или более остатков, а также вставки внутри последовательности одного или некоторого количества аминокислотных остатков. Примеры вставок включают CD28-антигенсвязывающие молекулы со слиянием в N- или С-конце с полипептидом, который увеличивает сывороточное время полужизни CD28-антигенсвязывающих молекул.
В определенных вариантах осуществления предложенные в данном документе CD28-антигенсвязывающие молекулы изменены с целью повышения или снижения степени гликозилирования антитела. Варианты молекул по гликозилированию удобно получать, изменяя аминокислотную последовательность так, чтобы создать или удалить один или более сайтов гликозилирования. Если агонистическая ICOS-связывающая молекула содержит Fc-домен, можно изменять присоединенный к ней углевод. Нативные антитела, вырабатываемые клетками млекопитающих, обычно содержат разветвленный биантеннарный олигосахарид, который обычно присоединен посредством N-связи к Asn297 СН2-домена Fc-области. Смотрите, например, Wright et al. TIBTECH 15:26-32 (1997). Олигосахарид может включать различные углеводы, например, маннозу, N-ацетилглюкозамин (GlcNAc), галактозу и сиаловую кислоту, а также фукозу, присоединенную к GlcNAc в «стволе» биантеннарной олигосахаридной структуры. В некоторых вариантах осуществления модификации олигосахарида в агонистических ICOS-связывающих молекулах можно осуществлять с целью создания вариантов с определенными улучшенными свойствами. В одном аспекте предложены варианты агонистических ICOS-связывающих молекул, содержащие углеводную структуру с недостаточным количеством фукозы, присоединенной (напрямую или не напрямую) к Fc-области. Такие варианты по фукозилированию могут иметь улучшенную функцию АЗКЦ, смотрите, например, публикации патентов США №№ US 2003/0157108 (Presta, L.) или US 2004/0093621 (Kyowa Hakko Kogyo Co., Ltd). Дополнительные варианты CD28-антигенсвязывающих молекул по изобретению включают варианты с разделенными олигосахаридами, например, в которых биантеннарный олигосахарид, присоединенный к Fc-области, разделен надвое GlcNAc. Такие варианты имеют сниженное фукозилирование и/или улучшенную функцию АЗКЦ, смотрите, например, WO 2003/011878 (Jean-Mairet et al.); патент США №6602684 (Umana et al.); и US 2005/0123546 (Umana et al.). Также предложены варианты с по меньшей мере одним остатком галактозы в олигосахариде, присоединенном к Fc-области. Такие варианты антител могут иметь улучшенную функцию КЗЦ и описаны, например, в WO 1997/30087 (Patel et al.); WO 1998/58964 (Raju, S.); и WO 1999/22764 (Raju, S.).
В определенных вариантах осуществления может быть желательно создавать цистеин-сконструированные варианты CD28-антигенсвязывающих молекул по изобретению, например, «thioMAb,» в которых один или более остатков молекулы замещены остатками цистеина. В конкретных вариантах осуществления замещенные остатки находятся в доступных сайтах молекулы. За счет замены этих остатков цистеином реакционноспособные тиольные группы располагаются в доступных сайтах антитела и могут быть использованы для конъюгации антитела с другими фрагментами, такими как лекарственные фрагменты или фрагменты линкер лекарственное средство, для создания иммуноконъюгата. В определенных вариантах осуществления цистеином может быть замещен любой из следующих остатков: V205 (нумерация Kabat) легкой цепи; A118 (нумерация EU) тяжелой цепи; и S400 (нумерация EU) Fc-области тяжелой цепи. Цистеин-сконструированные антигенсвязывающие молекулы можно создавать, как описано, например, в патенте США №7521541.
В определенных аспектах предложенные в данном документе CD28-антигенсвязывающие молекулы могут быть дополнительно модифицированы так, чтобы содержать дополнительные небелковые фрагменты, которые известны в данной области техники и легко доступны. Фрагменты, подходящие для дериватизации антитела, включают, но не ограничиваются этим, водорастворимые полимеры. Неограничивающие примеры водорастворимых полимеров включают, но не ограничиваются этим, полиэтиленгликоль (ПЭГ), сополимеры этиленгликоля/пропиленгликоля, карбоксиметилцеллюлозу, декстран, поливиниловый спирт, поливинилпирролидон, поли-1,3-диоксолан, поли-1,3,6-триоксан, сополимер этилена/малеинового ангидрида, полиаминокислоты (гомополимеры или случайные сополимеры) и декстран или поли(п-винилпирролидон)полиэтиленгликоль, гомополимеры пропропиленгликоля, сополимеры пропропиленоксида/этиленоксида, полиоксиэтилированные полиолы (например, глицерин), поливиниловый спирт и их смеси. Пропиональдегид полиэтиленгликоля может иметь преимущества при производстве из-за его стабильности в воде. Полимер может иметь любую молекулярную массу и может быть разветвленным или неразветвленным. Количество полимеров, прикрепленных к антителу, может варьироваться, и если прикреплено более одного полимера, они могут представлять собой одинаковые или различные молекулы. В целом, количество и/или тип полимеров, применяемых для дериватизации, можно определить на основании соображений, которые включают, но не ограничиваются этим, конкретные свойства или функции антитела, которые нужно улучшить, то, будет ли производное антитела применяться в терапии в определенных условиях и т.д. В другом аспекте предложены конъюгаты антитела и небелкового фрагмента, которые могут избирательно нагреваться под воздействием излучения. В одном варианте осуществления небелковый фрагмент представляет собой углеродную нанотрубку (Kam, N.W. et al., Proc. Nail. Acad. Sci. USA 102 (2005) 11600-11605).
Излучение может иметь любую длину волны и включает, но не ограничивается этим, длины волн, которые не вредят обычным клеткам, но которые нагревают небелковый фрагмент до температуры, при которой погибают клетки, расположенные вблизи конъюгата антитело - небелковый фрагмент. В другом аспекте можно получать иммуноконъюгаты предложенных в данном документе CD28-антигенсвязывающих молекул. «Иммуноконъюгат» представляет собой антитело; конъюгированное с одной или более гетерологичными молекулами, включая, но не ограничиваясь этим, цитотоксический агент.
Термин «полинуклеотид» относится к выделенной молекуле или конструкции нуклеиновой кислоты, например, матричной РНК (мРНК), вирусной РНК или плазмидной ДНК (пДНК). Полинуклеотид может содержать традиционную фосфодиэфирную связь или нетрадиционную связь (например, амидную связь, такую, как встречается в пептидных нуклеиновых кислотах (ПНК)). Термин «молекула нуклеиновой кислоты» относится к любым одному или более сегментам нуклеиновой кислоты, например, фрагментам ДНК или РНК, присутствующим в полинуклеотиде. Каждый нуклеотид состоит из основания, в частности, пуринового или пиримидинового основания (т.е. цитозина (С), гуанина (G), аденина (А), тимина (Т) или урацила (U)), сахара (т.е. дезоксирибозы или рибозы) и фосфатной группы. Часто молекула нуклеиновой кислоты описана по последовательности оснований, при этом указанные основания представляют первичную структуру (линейную структуру) молекулы нуклеиновой кислоты. Последовательность оснований, как правило, представлена от 5' к 3'. В данном документе термин молекула нуклеиновой кислоты охватывает дезоксирибонуклеиновую кислоту (ДНК), включая, например, комплементарную ДНК (кДНК) и геномную ДНК, рибонуклеиновую кислоту (РНК), в частности, матричную РНК (мРНК), синтетические формы ДНК и РНК и смешанные полимеры, содержащие две или более из этих молекул. Молекула нуклеиновой кислоты может быть линейной или кольцевой. Кроме того, термин молекула нуклеиновой кислоты включает как смысловые, так и антисмысловые цепи, а также одноцепочечные и двухцепочечные формы. Помимо этого, описанная в данном документе молекула нуклеиновой кислоты может содержать нуклеотиды природного и неприродного происхождения. Примеры нуклеотидов неприродного происхождения включают модифицированные нуклеотидные основания с дериватизированными сахарами, или фосфатными остовными связями, или химически модифицированными остатками. Молекулы нуклеиновых кислот также включают молекулы ДНК и РНК, подходящие в качестве вектора для управления экспрессией антитела по изобретению in vitro и/или in vivo, например, в организме хозяина или пациента. Такие ДНК (например, кДНК) или РНК (например, мРНК) векторы могут быть немодифицированными или модифицированными. Например, мРНК может быть химически модифицирована для повышения стабильности РНК-вектора и/или экспрессии кодируемой молекулы так, чтобы мРНК можно было вводить субъекту для генерации антител in vivo (смотрите, например, Stadler et al. (2017) Nature Medicine 23:815-817 или ЕР 2 101 823 B1).
Под «выделенными» молекулой нуклеиновой кислоты или полинуклеотидом подразумевается молекула нуклеиновой кислоты, ДНК или РНК, которая была удалена из своего нативного окружения. Например, рекомбинантный полинуклеотид, кодирующий полипептид, содержащийся в векторе, считается выделенным в целях настоящего изобретения. Дополнительные примеры выделенного полинуклеотида включают рекомбинантные полинуклеотиды, находящиеся в гетерологичных клетках-хозяевах, или очищенные (частично или по существу) полинуклеотиды в растворе. Выделенный полинуклеотид включает молекулу полинуклеотида, содержащуюся в клетках, которые обычно содержат эту молекулу полинуклеотида, но при этом молекула полинуклеотида присутствует внехромосомно или в хромосомном положении, которое отличается от ее природного хромосомного положения. Выделенные молекулы РНК включают in vivo или in vitro РНК-транскрипты по настоящему изобретению, а также положительные и отрицательные формы цепей и двухцепочечные формы. Выделенные полинуклеотиды или нуклеиновые кислоты в соответствии с настоящим изобретением дополнительно включают такие молекулы, полученные синтетически. Дополнительно полинуклеотид или нуклеиновая кислота могут представлять собой или включать регуляторный элемент, такой как промотор, сайт связывания рибосомы или терминатор транскрипции.
Под нуклеиновой кислотой или полинуклеотидом, имеющими нуклеотидную последовательность, по меньшей мере, например, на 95% «идентичную» референсной последовательности по настоящему изобретению, подразумевается, что нуклеотидная последовательность полинуклеотида идентична референсной последовательности за исключением того, что полинуклеотидная последовательность может включать до пяти точечных мутаций на каждые 100 нуклеотидов референсной нуклеотидной последовательности. Другими словами; чтобы получить полинуклеотид, имеющий нуклеотидную последовательность, по меньшей мера на 95% идентичную референсной нуклеотидной последовательности, до 5% нуклеотидов в референсной последовательности могут быть удалены или замещены другими нуклеотидами или в референсную последовательность может быть вставлено число нуклеотидов, составляющее до 5% от общего числа нуклеотидов в референсной последовательности. Эти изменения референсной последовательности могут находиться в 5' или 3' концевых позициях референсной нуклеотидной последовательности или в любом месте между этими концевыми позициями, размещаясь по отдельности между остатками в референсной последовательности или одной или более непрерывными группами в референсной последовательности. На практике определение того, что любая конкретная полинуклеотидная последовательность является по меньшей мере на 80%, 85%, 90%, 95%, 96%, 97%, 98% или 99% идентичной нуклеотидной последовательности по настоящему изобретению, можно осуществлять традиционным способом, используя известные компьютерные программы, такие как те, которые обсуждаются выше для полипептидов (например, ALIGN-2).
Термин «экспрессионная кассета» относится к полинуклеотиду, созданному рекомбинантным или синтетическим способом, с рядом определенных элементов нуклеиновой кислоты, которые обеспечивают возможность транскрипции конкретной нуклеиновой кислоты в целевой клетке. Рекомбинантная экспрессионная кассета может быть включена в плазмиду, хромосому, митохондриальную ДНК, пластидную ДНК, вирус или фрагмент нуклеиновой кислоты. Как правило, рекомбинантная экспрессионная кассета, как часть экспрессионного вектора, содержит, помимо других последовательностей, последовательность нуклеиновой кислоты, предназначенную для транскрипции, и промотор. В определенных вариантах осуществления экспрессионная кассета по изобретению содержит полинуклеотидные последовательности, которые кодируют биспецифические антигенсвязывающие молекулы по изобретению или их фрагменты.
Термины «вектор» или «экспрессионный вектор» являются синонимами с «экспрессионной конструкцией» и обозначают молекулу ДНК, которую используют для внесения и управления экспрессией конкретного гена, с которым она функционально связана, в целевой клетке. Этот термин включает вектор в виде самореплицирующейся структуры нуклеиновой кислоты, а также вектор, включенный в геном клетки-хозяина, в которую его внесли. Экспрессионный вектор по настоящему изобретению содержит экспрессионную кассету. Экспрессионные векторы делают возможной транскрипцию больших количеств стабильной мРНК. После того, как экспрессионный вектор оказывается в целевой клетке, клеточная машинерия транскрипции и/или трансляции вырабатывает молекулу рибонуклеиновой кислоты или белок, кодируемые геном. В одном варианте осуществления экспрессионный вектор по изобретению содержит экспрессионную кассету, которая содержит полинуклеотидные последовательности, которые кодируют биспецифические антигенсвязывающие молекулы по изобретению или их фрагменты.
Термины «клетка-хозяин», «линия клеток-хозяев» и «культура клеток-хозяев» используются взаимозаменяемо и относятся к клеткам, в которые была внесена экзогенная нуклеиновая кислота, включая потомство таких клеток. Клетки-хозяева включают «транс фор манто в» и «трансформированные клетки», которые включают первично трансформированные клетки и полученное от них потомство вне зависимости от числа пассажей. Потомство может не быть полностью идентичным родительской клетке по содержанию нуклеиновой кислоты и может содержать мутации. Сюда включено мутантное потомство, которое имеет такую же функцию или биологическую активность, в отношении которых проводится скрининг или отбор изначально трансформированных клеток. Клетка-хозяин принадлежит любому типу клеточной системы, который можно использовать для создания биспецифических антигенсвязывающих молекул по настоящему изобретению. Клетки-хозяева включают культивируемые клетки, например, культивируемые клетки млекопитающих, такие как клетки СНО, клетки BHK, клетки NSO, клетки SP2/0, клетки миеломы YO, клетки миеломы мышей Р3Х63, клетки PER, клетки PER.C6 или клетки гибридомы, клетки дрожжей, клетки насекомых и клетки растений, если называть только некоторые, но также клетки, принадлежащие трансгенному животному, трансгенному растению или культивируемой растительной или животной ткани.
«Эффективное количество» агента относится к количеству, необходимому для физиологического изменения в клетке или ткани, в которую его вводят.
«Терапевтически эффективное количество» агента, например, фармацевтической композиции, относится к количеству, эффективному в дозировках и в течение периодов времени, необходимых для достижения желательного терапевтического или профилактического результата. Терапевтически эффективное количество агента, например, устраняет, снижает, замедляет, минимизирует или предотвращает нежелательные явления заболевания.
«Индивид» или «субъект» представляет собой млекопитающее. Млекопитающие включают, но не ограничиваются этим, одомашненных животных (например, коров, овец, кошек, собак и лошадей), приматов (например, людей и отличных от человека приматов, таких, как обезьяны), кроликов и грызунов (например, мышей и крыс). В частности, индивид или субъект представляет собой человека.
Термин «фармацевтическая композиция» относится к препарату, который находится в форме, обеспечивающей эффективность биологической активности содержащегося в нем активного ингредиента, и который не содержит дополнительные компоненты, которые являются неприемлемо токсичными для субъекта, которому будет введен состав.
«Фармацевтически приемлемый эксципиент» относится к ингредиенту в фармацевтической композиции, отличному от активного ингредиента, который нетоксичен для субъекта. Фармацевтически приемлемый эксципиент включает, но не ограничивается этим, буфер, вспомогательное вещество, стабилизатор или консервант.
Термин «вкладыш в упаковку» используется для обозначения инструкций, обычно включаемых в коммерческие упаковки терапевтических продуктов, которые содержат информацию о показаниях, применении, дозировке, введении, комбинированной терапии, противопоказаниях и/или предостережениях относительно применения таких терапевтических продуктов.
В контексте данного документа термин «лечение» (и его грамматические варианты, такие как «лечить» или «лечащий») относится к клиническому вмешательству с целью изменения естественного течения заболевания индивида, подлежащего лечению, и может проводиться как для профилактики, так и в течении клинической патологии. Необходимые эффекты лечения включают, но не ограничиваются этим, предотвращение появления или повторного появления заболевания, смягчение симптомов, уменьшение каких-либо прямых или косвенных патологических последствий заболевания, предотвращение метастазирования, снижение скорости прогрессирования заболевания, уменьшение интенсивности или временное облегчение болезненного состояния и ремиссию или улучшение прогноза. В некоторых вариантах осуществления молекулы по изобретению применяют для задержки развития заболевания или замедления прогрессирования заболевания.
Термины «комбинированное лечение» или «совместное введение», упоминаемые в данном документе, охватывают комбинированное введение (при котором два или более терапевтических агентов включены в один или в разные составы) и раздельное введение, в случае которого введение антитела по данному изобретению может происходить до, одновременно и/или после введения дополнительного терапевтического агента или агентов, предпочтительно антитела или антител.
Под «В-клеточным пролиферативным нарушением» подразумевается заболевание, при котором число В-клеток у пациента повышено по сравнению с числом В-клеток у здорового субъекта и, в частности, при котором повышение числа В-клеток является признаком заболевания.
Термин «гематологический рак» относится к или описывает физиологическое состояние у млекопитающих, которое, как правило, характеризуется нерегулируемым ростом/пролиферацией клеток. Таким образом, в контексте данного документа термин рак относится к пролиферативным нарушениям, таким как карцинома, лимфомы (например, ходжкинская и неходжкинская лимфома), бластома, саркома и лейкоз. В частности, термин рак относится к В-клеточному пролиферативному нарушению. В одном аспекте рак выбран из группы, состоящей из неходжкинской лимфомы (НХЛ), острого лимфоцитарного лейкоза (ОЛЛ), хронического лимфоцитарного лейкоза (ХЛЛ), диффузной В-крупноклеточной лимфомы (ДВКЛ), филликулярной лимфомц (ФЛ), мантийноклеточной лимфому (МКЛ), лимфомы маргинальной зоны (ЛМЗ), множественной миеломы (ММ) и лимфомы Ходжкина (ЛХ).
Термин «рак» относится к или описывает физиологическое состояние у млекопитающих, которое, как правило, характеризуется нерегулируемым ростом/пролиферацией клеток. Таким образом, в контексте данного документа термин рак относится к пролиферативным нарушениям, таким как карцинома, лимфомы (например, ходжкинская и неходжкинская лимфома), бластома, саркома и лейкоз. В частности, термин рак включает лимфоцитарные лейкозы, рак легкого, немелкоклеточный рак легких (НМРЛ), рак легкого из бронхиолоальвеолярных клеток, рак кости, рак поджелудочной железы, рак кожи, рак головы и шеи, кожную или интраокулярную меланому, рак матки, рак яичника, ректальный рак, рак анальной области, рак желудка, рак толстой кишки, рак молочной железы, рак матки, карциному фаллопиевых труб, карциному эндометрия, карциному шейки матки, карциному вагины, карциному вульвы, болезнь Ходжкина, рак пищевода, рак тонкого кишечника, рак эндокринной системы, рак щитовидной железы, рак паращитовидной железы, рак надпочечника, саркому мягкой ткани, рак уретры, рак полового члена, рак предстательной железы, рак мочевого пузыря, рак почки или мочеточника, почечно-клеточную карциному, карциному почечной лоханки, мезотелиому, печеночноклеточный рак, рак желчного пузыря, новообразования центральной нервной системы (ЦНС), опухоли оси позвоночника, глиому ствола головного мозга, мультиформную глиобластому, астроцитомы, шванномы, эпендимомы, медуллобластомы, менингиомы, плоскоклеточные карциномы, аденому гипофиза и саркому Юинга, включая рефракторные версии любого из вышеперечисленных видов рака или комбинации одного или более из вышеперечисленных видов рака. В одном аспекте рак представляет собой солидную опухоль. В другом аспекте рак представляет собой гематологический рак, в частности лейкоз, конкретнее острый лимфобластный лейкоз (ОЛЛ) или острый миелогенный лейкоз (ОМЛ).
Биспецифические агонистические CD28-антигенсвязывающие молекулы по изобретению
В изобретении предложены новые биспецифические агонистические CD28-антигенсвязывающие молекулы с исключительно преимущественными свойствами, такими как технологичность, стабильность, аффинность связывания, биологическая активность, эффективность нацеливания, сниженная токсичность, расширенный диапазон дозировок, которые можно вводить пациенту, и, следовательно, возможно повышенная эффективность. Новые биспецифические агонистические CD28-антигенсвязывающие молекулы содержат Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию (Fc-сайленсинг) и, таким образом, предотвращается перекрестное связывание посредством Fc-рецепторов. Вместо этого они содержат по меньшей мере один антигенсвязывающий домен, способный к специфическому связыванию с опухолеассоциированным антигеном, таким как белок активации фибробластов (FAP) или карциноэмбриональный антиген (СЕА), что приводит к перекрестному связыванию в месте опухоли. Таким образом, обеспечивается активация опухолеспецифических Т-клеток.
В данном документе предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула с одновалентным связыванием с CD28, содержащая
(а) один антигенсвязывающий домен, способный к специфическому связыванию с CD28,
(б) по меньшей мере один антигенсвязывающий домен, способный к специфическому связыванию с опухолеассоциированным антигеном, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула по определению ниже в данном документе, в которой Fc-домен принадлежит IgG, в частности представляет собой Fc-домен IgG1 или Fc-домен IgG4. В одном конкретном аспекте Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, представляет собой Fc-домен IgG1. Fc-домен содержит одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или снижает или устраняет эффекторную функцию. В одном аспекте Fc-домен содержит аминокислотные замены L234A и L235A (нумерация в соответствии с индексом EU по Kabat). В одном аспекте Fc-домен принадлежит подклассу IgG1 человека и содержит аминокислотные мутации L234A, L235A и P329G (нумерация в соответствии с индексом EU по Kabat). В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, причем антигенсвязывающая молекула содержит Ес-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, в котором первая субъединица содержит аминокислотную последовательность SEQ ID NO: 176, а вторая субъединица содержит аминокислотную последовательность SEQ ID NO: 177.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула по определению выше в данном документе, в которой по меньшей мере один антигенсвязывающий домен, способный к специфическому связыванию с CD28, содержит (i) вариабельную область тяжелой цепи (VHCD28), содержащую определяющую комплементарность область тяжелой цепи CDR-H1 с SEQ ID NO: 20, CDR-H2 с SEQ ID NO: 21 и CDR-Н3 с SEQ ID NO: 22, и вариабельную область легкой цепи (VLCD28), содержащую определяющую комплементарность область легкой цепи CDR-L1 с SEQ ID NO: 23, CDR-L2 с SEQ ID NO: 24 и CDR-L3 с SEQ ID NO: 25; или
(ii) вариабельную область тяжелой цепи (VHCD28), содержащую CDR-H1 с SEQ ID NO: 36, CDR-H2 с SEQ ID NO: 37 и CDR-Н3 с SEQ ID NO: 38, и вариабельную область легкой цепи (VLCD28), содержащую CDR-L1 с SEQ ID NO: 39, CDR-L2 с SEQ ID NO: 40 и CDR-L3 с SEQ ID NO: 41.
В одном аспекте антигенсвязывающий домен, способный к специфическому связыванию с CD28, биспецифической агонистической CD28-антигенсвязывающей молекулы содержит вариабельную область тяжелой цепи (VHCD28), содержащую CDR-H1 с SEQ ID NO: 36, CDR-H2 с SEQ ID NO: 37 и CDR-H3 с SEQ ID NO: 38, и вариабельную область легкой цепи (VLCD28), содержащую CDR-L1 с SEQ ID NO: 39, CDR-L2 с SEQ ID NO: 40 и CDR-L3 с SEQ ID NO: 41.
В одном аспекте антигенсвязывающий домен, способный к специфическому связыванию с CD28, биспецифической агонистической CD28-антигенсвязывающей молекулы содержит вариабельную область тяжелой цепи (VHCD28), содержащую CDR-H1 с SEQ ID NO: 20, CDR-H2 с SEQ ID NO: 21 и CDR-H3 с SEQ ID NO: 22, и вариабельную область легкой цепи (VLCD28), содержащую CDR-L1 с SEQ ID NO: 23, CDR-L2 с SEQ ID NO: 24 и CDR-L3 с SEQ ID NO: 25.
Кроме того, предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула по определению выше в данном документе, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD28, содержит вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 26, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 27.
В другом аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD28, содержит
(а) CDR вариабельной области тяжелой цепи (VHCD28), содержащей аминокислотную последовательность SEQ ID NO: 47, и CDR вариабельной области легкой цепи (VLCD28), содержащей аминокислотную последовательность SEQ ID NO: 54, или
(б) CDR вариабельной области тяжелой цепи (VHCD28), содержащей аминокислотную последовательность SEQ ID NO: 47, и CDR вариабельной области легкой цепи (VLCD28), содержащей аминокислотную последовательность SEQ ID NO: 27, или
(в) CDR вариабельной области тяжелой цепи (VHCD28), содержащей аминокислотную последовательность SEQ ID NO: 51, и CDR вариабельной области легкой цепи (VLCD28), содержащей аминокислотную последовательность SEQ ID NO: 61, или
(г) CDR вариабельной области тяжелой цепи (VHCD28), содержащей аминокислотную последовательность SEQ ID NO: 46, и CDR вариабельной области легкой цепи (VLCD28), содержащей аминокислотную последовательность SEQ ID NO: 53, или
(д) CDR вариабельной области тяжелой цепи (VHCD28), содержащей аминокислотную последовательность SEQ ID NO: 46, и CDR вариабельной области легкой цепи (VLCD28), содержащей аминокислотную последовательность SEQ ID NO: 54, или
(е) CDR вариабельной области тяжелой цепи (VHCD28), содержащей аминокислотную последовательность SEQ ID NO: 46; и CDR вариабельной области легкой цепи (VLCD28), содержащей аминокислотную последовательность SEQ ID NO: 59, или
(ж) CDR вариабельной области тяжелой цепи (VHCD28), содержащей аминокислотную последовательность SEQ ID NO: 46, и CDR вариабельной области легкой цепи (VLCD28), содержащей аминокислотную последовательность SEQ ID NO: 27, или
(и) CDR вариабельной области тяжелой цепи (VHCD28), содержащей аминокислотную последовательность SEQ ID NO: 43, и CDR вариабельной области легкой цепи (VLCD28), содержащей аминокислотную последовательность SEQ ID NO: 27, или
(к) CDR вариабельной области тяжелой цепи (VHCD28), содержащей аминокислотную последовательность SEQ ID NO: 42, и CDR вариабельной области легкой цепи (VLCD28), содержащей аминокислотную последовательность SEQ ID NO: 53, или
(л) CDR вариабельной области тяжелой цепи (VHCD28), содержащей аминокислотную последовательность SEQ ID NO: 42, и CDR вариабельной области легкой цепи (VLCD28), содержащей аминокислотную последовательность SEQ ID NO: 59, или
(м) CDR вариабельной области тяжелой цепи (VHCD28), содержащей аминокислотную последовательность SEQ ID NO: 42, и CDR вариабельной области легкой цепи (VLCD28), содержащей аминокислотную последовательность SEQ ID NO: 27.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD28, содержит CDR вариабельной области тяжелой цепи (VHCD28), содержащей аминокислотную последовательность SEQ ID NO: 47, и CDR вариабельной области легкой цепи (VLCD28), содержащей аминокислотную последовательность SEQ ID NO: 54. В одном аспекте антигенсвязывающий домен, способный к специфическому связыванию с CD28, биспецифической агонистической CD28-антигенсвязывающей молекулы содержит вариабельную область тяжелой цепи (VHCD28), содержащую CDR-H1 с SEQ ID NO: 489, CDR-H2 с SEQ ID NO: 490 и CDR-H3 с SEQ ID NO: 491, и вариабельную область легкой цепи (VLCD28), содержащую CDR-L1 с SEQ ID NO: 492, CDR-L2 с SEQ ID NO: 493 и CDR-L3 с SEQ ID NO: 494.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD28, содержит CDR вариабельной области тяжелой цепи (VHCD28), содержащей аминокислотную последовательность SEQ ID NO: 46, и CDR вариабельной области легкой цепи (VLCD28), содержащей аминокислотную последовательность SEQ ID NO: 53. В одном аспекте антигенсвязывающий домен, способный к специфическому связыванию с CD28, биспецифической агонистической CD28-антигенсвязывающей молекулы содержит вариабельную область тяжелой цепи (VHCD28), содержащую CDR-H1 с SEQ ID NO: 495, CDR-H2 с SEQ ID NO: 496 и CDR-H3 с SEQ ID NO: 497, и вариабельную область легкой цепи (VLCD28), содержащую CDR-L1 с SEQ ID NO: 498, CDR-L2 с SEQ ID NO: 499 и CDR-L3 с SEQ ID NO: 500.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD28, содержит CDR вариабельной области тяжелой цепи (VHCD28), содержащей аминокислотную последовательность SEQ ID NO: 42, и CDR вариабельной области легкой цепи (VLCD28), содержащей аминокислотную последовательность SEQ ID NO: 27. В одном аспекте антигенсвязывающий домен, способный к специфическому связыванию с CD28, биспецифической агонистической CD28-антигенсвязывающей молекулы содержит вариабельную область тяжелой цепи (VHCD28), содержащую CDR-H1 с SEQ ID NO: 501, CDR-H2 с SEQ ID NO: 502 и CDR-H3 с SEQ ID NO: 503, и вариабельную область легкой цепи (VLCD28), содержащую CDR-L1 с SEQ ID NO: 504, CDR-L2 с SEQ ID NO: 505 и CDR-L3 с SEQ ID NO: 506.
В дополнительном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD28, содержит вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 42, SEQ ID NO: 43, SEQ ID NO: 44, SEQ ID NO: 45, SEQ ID NO: 46, SEQ ID NO: 47, SEQ ID NO: 48, SEQ ID NO: 49, SEQ ID NO: 50 и SEQ ID NO: 51, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 27, SEQ ID NO: 52, SEQ ID NO: 53, SEQ ID NO: 54, SEQ ID NO: 55, SEQ ID NO: 56, SEQ ID NO: 57, SEQ ID NO: 58, SEQ ID NO: 59, SEQ ID NO: 60 и SEQ ID NO: 61.
В другом аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD28, содержит
(а) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 47, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 54, или
(б) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 47, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 27, или
(в) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 51, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 61, или
(г) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 53, или
(д) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 54, или
(е) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 59, или
(ж) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 27, или
(и) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 43, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 27, или
(к) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 42, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 53, или
(л) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 42, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 59, или
(м) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 42, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 27.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD28, связывается с CD28 со сниженной аффинностью по сравнению с антигенсвязывающим доменом, содержащим вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 26, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 27. Аффинность измеряют методом проточной цитометрии как связывание с клетками СНО, экспрессирующими CD28. В одном аспекте антигенсвязывающий домен, способный к специфическому связыванию с CD28, связывается с CD28 со сниженной аффинностью по сравнению с антигенсвязывающим доменом, содержащим вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 26, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 27, содержит CDR-H1, CDR-H2 и CDR-H3 вариабельной области тяжелой цепи (VHCD28), содержащей аминокислотную последовательность SEQ ID NO: 47, и CDR-L1, CDR-L2 и CDR-L3 вариабельной области легкой цепи (VLCD28), содержащей аминокислотную последовательность SEQ ID NO: 54. В одном аспекте антигенсвязывающий домен, способный к специфическому связыванию с CD28 со сниженной аффинностью по сравнению с антигенсвязывающим доменом, содержащим вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 26, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 27, содержит вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 47, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 54.
В одном конкретном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD28, содержит вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 47, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 54.
В другом конкретном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD28, содержит вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 53.
В дополнительном конкретном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD28, содержит вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 42, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 27.
Нацеленные на СЕА биспецифические агонистические CD28-антигенсвязывающие молекулы
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с опухолеассоциированным антигеном, представляет собой антигенсвязывающий домен, способный к специфическому связыванию с карциноэмбриональным антигеном (СЕА).
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, в которой по меньшей мере один антигенсвязывающий домен, способный к специфическому связыванию с СЕА, содержит (i) вариабельную область тяжелой цепи (VHCEA), содержащую CDR-H1, содержащую аминокислотную последовательность SEQ ID NO: 188, CDR-H2, содержащую аминокислотную последовательность SEQ ID NO: 189, и CDR-H3, содержащую аминокислотную последовательность SEQ ID NO: 190, и вариабельную область легкой цепи (VHCEA), содержащую CDR-L1, содержащую аминокислотную последовательность SEQ ID NO: 191, CDR-L2, содержащую аминокислотную последовательность SEQ ID NO: 192, и CDR-L3, содержащую аминокислотную последовательность SEQ ID NO: 193; или (ii) вариабельную область тяжелой цепи (VHCEA), содержащую CDR-H1, содержащую аминокислотную последовательность SEQ ID NO: 180, CDR-H2, содержащую аминокислотную последовательность SEQ ID NO: 181, и CDR-H3, содержащую аминокислотную последовательность SEQ ID NO: 182, и вариабельную область легкой цепи (VHCEA), содержащую CDR-L1, содержащую аминокислотную последовательность SEQ ID NO: 183, CDR-L2, содержащую аминокислотную последовательность SEQ ID NO: 184, и CDR-L3, содержащую аминокислотную последовательность SEQ ID NO: 185; или
(iii) вариабельную область тяжелой цепи (VHCEA), содержащую CDR-H1, содержащую аминокислотную последовательность SEQ ID NO: 127, CDR-H2, содержащую аминокислотную последовательность SEQ ID NO: 128, и CDR-H3, содержащую аминокислотную последовательность SEQ ID NO: 129, и вариабельную область легкой цепи (VLCEA), содержащую CDR-L1, содержащую аминокислотную последовательность SEQ ID NO: 130, CDR-L2, содержащую аминокислотную последовательность SEQ ID NO: 131, и CDR-L3, содержащую аминокислотную последовательность SEQ ID NO: 132, или (iv) вариабельную область тяжелой цепи (VHCEA), содержащую CDR-H1, содержащую аминокислотную последовательность SEQ ID NO: 507, CDR-H2, содержащую аминокислотную последовательность SEQ ID NO: 508, и CDR-H3, содержащую аминокислотную последовательность SEQ ID NO: 509, и вариабельную область легкой цепи (vlcea), содержащую CDR-L1, содержащую аминокислотную последовательность SEQ ID NO: 510, CDR-L2, содержащую аминокислотную последовательность SEQ ID NO: 511, и CDR-L3, содержащую аминокислотную последовательность SEQ ID NO: 512.
В одном конкретном аспекте антигенсвязывающий домен, способный к специфическому связыванию с СЕА, содержит вариабельную область тяжелой цепи (VHCEA), содержащую CDR-H1, содержащую аминокислотную последовательность SEQ ID NO: 188, CDR-H2, содержащую аминокислотную последовательность SEQ ID NO: 189, и CDR-H3, содержащую аминокислотную последовательность SEQ ID NO: 190, и вариабельную область легкой цепи (vlcea), содержащую CDR-L1, содержащую аминокислотную последовательность SEQ ID NO: 191, CDR-L2, содержащую аминокислотную последовательность SEQ ID NO: 192, и CDR-L3, содержащую аминокислотную последовательность SEQ ID NO: 193.
В частности, антигенсвязывающий домен, способный к специфическому связыванию с СЕА, содержит вариабельную область тяжелой цепи (VHCEA), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 133, и вариабельную область легкой цепи (VLCEA), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 134. В одном аспекте антигенсвязывающий домен, способный к специфическому связыванию с СЕА, содержит вариабельную область тяжелой цепи (VHCEA), содержащую аминокислотную последовательность SEQ ID NO: 133, и вариабельную область легкой цепи (vlcea), содержащую аминокислотную последовательность SEQ ID NO: 134.
В другом аспекте антигенсвязывающий домен, способный к специфическому связыванию с СЕА, содержит вариабельную область тяжелой цепи (VHCEA), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 186, и вариабельную область легкой цепи (vlcea), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 187. В одном аспекте антигенсвязывающий домен, способный к специфическому связыванию с СЕА, содержит вариабельную область тяжелой цепи (VHCEA), содержащую аминокислотную последовательность SEQ ID NO: 186, и вариабельную область легкой цепи (vlcea), содержащую аминокислотную последовательность SEQ ID NO: 187.
В другом аспекте антигенсвязывающий домен, способный к специфическому связыванию с СЕА, содержит вариабельную область тяжелой цепи (VHCEA), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 513, и вариабельную область легкой цепи (vlcea), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 514. В одном аспекте антигенсвязывающий домен, способный к специфическому связыванию с СЕА, содержит вариабельную область тяжелой цепи (VHCEA), содержащую аминокислотную последовательность SEQ ID NO: 513, и вариабельную область легкой цепи (vlcea), содержащую аминокислотную последовательность SEQ ID NO: 514.
В другом аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с СЕА, содержит
(а) вариабельную область тяжелой цепи (VHCEA), содержащую аминокислотную последовательность SEQ ID NO: 194, и вариабельную область легкой цепи (vlcea), содержащую аминокислотную последовательность SEQ ID NO: 195. или
(б) вариабельную область тяжелой цепи (VHCEA), содержащую аминокислотную последовательность SEQ ID NO: 196, и вариабельную область легкой цепи (vlcea), содержащую аминокислотную последовательность SEQ ID NO: 197, или
(в) вариабельную область тяжелой цепи (VHCEA), содержащую аминокислотную последовательность SEQ ID NO: 198, и вариабельную область легкой цепи (vlcea), содержащую аминокислотную последовательность SEQ ID NO: 199, или
(г) вариабельную область тяжелой цепи (VHCEA), содержащую аминокислотную последовательность SEQ ID NO: 200, и вариабельную область легкой цепи (vlcea), содержащую аминокислотную последовательность SEQ ID NO: 201, или
(д) вариабельную область тяжелой цепи (VHCEA), содержащую аминокислотную последовательность SEQ ID NO: 202, и вариабельную область легкой цепи (vlcea), содержащую аминокислотную последовательность SEQ ID NO: 203, или
(е) вариабельную область тяжелой цепи (VHCEA), содержащую аминокислотную последовательность SEQ ID NO: 204, и вариабельную область легкой цепи (vlcea), содержащую аминокислотную последовательность SEQ ID NO: 205, или
(ж) вариабельную область тяжелой цепи (VHCEA), содержащую аминокислотную последовательность SEQ ID NO: 206, и вариабельную область легкой цепи (vlcea), содержащую аминокислотную последовательность SEQ ID NO: 207, или
(и) вариабельную область тяжелой цепи (VHCEA), содержащую аминокислотную последовательность SEQ ID NO: 208, и вариабельную область легкой цепи (vlcea), содержащую аминокислотную последовательность SEQ ID NO: 209, или
(к) вариабельную область тяжелой цепи (VHCEA), содержащую аминокислотную последовательность SEQ ID NO: 210, и вариабельную область легкой цепи (vlcea), содержащую аминокислотную последовательность SEQ ID NO: 211, или
(л) вариабельную область тяжелой цепи (VHCEA), содержащую аминокислотную последовательность SEQ ID NO: 212, и вариабельную область легкой цепи (vlcea), содержащую аминокислотную последовательность SEQ ID NO: 213.
В частности, антигенсвязывающий домен, способный к специфическому связыванию с СЕА, содержит вариабельную область тяжелой цепи (VHCEA), содержащую аминокислотную последовательность SEQ ID NO: 200, и вариабельную область легкой цепи (vlcea), содержащую аминокислотную последовательность SEQ ID NO: 201.
Нацеленные на FAP биспецифические агонистические CD28-антигенсвязывающие молекулы
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с опухолеассоциированным антигеном, представляет собой антигенсвязывающий домен, способный к специфическому связыванию с белком активации фибробластов (FAP).
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, в которой по меньшей мере один антигенсвязывающий домен, способный к специфическому связыванию с FAP, содержит
(а) вариабельную область тяжелой цепи (VHFAP), содержащую (i) CDR-H1, содержащую аминокислотную последовательность SEQ ID NO: 12, (ii) CDR-H2, содержащую аминокислотную последовательность SEQ ID NO: 13, и (iii) CDR-H3, содержащую аминокислотную последовательность SEQ ID NO: 14, и вариабельную область легкой цепи (vlfap), содержащую (iv) CDR-L1, содержащую аминокислотную последовательность SEQ ID NO: 15, (v) CDR-L2, содержащую аминокислотную последовательность SEQ ID NO: 16, и (vi) CDR-L3, содержащую аминокислотную последовательность SEQ ID NO: 17, или (б) вариабельную область тяжелой цепи (VHFAP), содержащую (i) CDR-H1, содержащую аминокислотную последовательность SEQ ID NO: 4, (ii) CDR-H2, содержащую аминокислотную последовательность SEQ ID NO: 5, и (iii) CDR-H3, содержащую аминокислотную последовательность SEQ ID NO: 6, и вариабельную область легкой цепи (vlfap), содержащую (iv) CDR-L1, содержащую аминокислотную последовательность SEQ ID NO: 7, (v) CDR-L2, содержащую аминокислотную последовательность SEQ ID NO: 8, и (vi) CDR-L3, содержащую аминокислотную последовательность SEQ ID NO: 9.
В частности, антигенсвязывающий домен, способный к специфическому связыванию с FAP, содержит вариабельную область тяжелой цепи (VHFAP), содержащую (i) CDR-H1, содержащую аминокислотную последовательность SEQ ID NO: 12, (ii) CDR-H2, содержащую аминокислотную последовательность SEQ ID NO: 13, и (iii) CDR-H3, содержащую аминокислотную последовательность SEQ ID NO: 14, и вариабельную область легкой цепи (vlfap), содержащую (iv) CDR-L1, содержащую аминокислотную последовательность SEQ ID NO: 15, (v) CDR-L2, содержащую аминокислотную последовательность SEQ ID NO: 16, и (vi) CDR-L3, содержащую аминокислотную последовательность SEQ ID NO: 17. В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с FAP, содержит (а) вариабельную область тяжелой цепи (VHFAP), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 18, и вариабельную область легкой цепи (vlfap), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 19, или (б) вариабельную область тяжелой цепи (VHFAP), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 10, и вариабельную область легкой цепи (vlfap), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 11. В частности, антигенсвязывающий домен, способный к специфическому связыванию с FAP, содержит вариабельную область тяжелой цепи (VHFAP), содержащую аминокислотную последовательность SEQ ID NO: 18, и вариабельную область легкой цепи (vlfap), содержащую аминокислотную последовательность SEQ ID NO: 19.
Нацеленные на ЕрСАМ биспецифические агонистические CD28-антигенсвязывающие молекулы
В другом аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с опухолеассоциированным антигеном, представляет собой антигенсвязывающий домен, способный к специфическому связыванию с молекулой адгезии эпителиальных клеток (ЕрСАМ).
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, в которой антигенсвязывающий домен, способный к специфическому связыванию с ЕрСАМ, содержит вариабельную область тяжелой цепи (VHEpCAM), содержащую (i) CDR-H1, содержащую аминокислотную последовательность SEQ ID NO: 515, (ii) CDR-H2, содержащую аминокислотную последовательность SEQ ID NO: 516, и (iii) CDR-H3, содержащую аминокислотную последовательность SEQ ID NO: 517, и вариабельную область легкой цепи (VLEpCAM), содержащую (iv) CDR-L1, содержащую аминокислотную последовательность SEQ ID NO: 518, (v) CDR-L2, содержащую аминокислотную последовательность SEQ ID NO: 519, и (vi) CDR-L3, содержащую аминокислотную последовательность SEQ ID NO: 520. В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с ЕрСАМ, содержит вариабельную область тяжелой цепи (VHEpCAM), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 521, и вариабельную область легкой цепи (VLEpCAM), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 522. В частности, антигенсвязывающий домен, способный к специфическому связыванию с ЕрСАМ, содержит вариабельную область тяжелой цепи (VHEpCAM), содержащую аминокислотную последовательность SEQ ID NO: 521, и вариабельную область легкой цепи (VLEpCAM), содержащую аминокислотную последовательность SEQ ID NO: 522.
Нацеленные на HER3 биспецифические агонистические CD28-антигенсвязывающие молекулы
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с опухолеассоциированным антигеном, представляет собой антигенсвязывающий домен, способный к специфическому связыванию с HER3.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, в которой антигенсвязывающий домен, способный к специфическому связыванию с HER3, содержит вариабельную область тяжелой цепи (VHHER3), содержащую (i) CDR-Н1, содержащую аминокислотную последовательность SEQ ID NO: 523, (ii) CDR-H2, содержащую аминокислотную последовательность SEQ ID NO: 524, и (iii) CDR-H3, содержащую аминокислотную последовательность SEQ ID NO: 525, и вариабельную область легкой цепи (VLHER3), содержащую (iv) CDR-L1, содержащую аминокислотную последовательность SEQ ID NO: 526, (v) CDR-L2, содержащую аминокислотную последовательность SEQ ID NO: 527, и (vi) CDR-L3, содержащую аминокислотную последовательность SEQ ID NO: 528. В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с HER3, содержит вариабельную область тяжелой цепи (VHHER3), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 529, и вариабельную область легкой цепи (VLHER3), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 530. В частности, антигенсвязывающий домен, способный к специфическому связыванию с HER3, содержит вариабельную область тяжелой цепи (VHHER3), содержащую аминокислотную последовательность SEQ ID NO: 529, и вариабельную область легкой цепи (VLHER3), содержащую аминокислотную последовательность SEQ ID NO: 530.
Нацеленные на CD30 биспецифические агонистические CD28-антигенсвязывающие молекулы
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с опухолеассоциированным антигеном, представляет собой антигенсвязывающий домен, способный к специфическому связыванию с CD30.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD30, содержит вариабельную область тяжелой цепи (VHCD30), содержащую (i) CDR-Н1, содержащую аминокислотную последовательность SEQ ID NO: 531, (ii) CDR-H2, содержащую аминокислотную последовательность SEQ ID NO: 532, и (iii) CDR-H3, содержащую аминокислотную последовательность SEQ ID NO: 533, и вариабельную область легкой цепи (VLCD30), содержащую (iv) CDR-L1, содержащую аминокислотную последовательность SEQ ID NO: 534, (v) CDR-L2, содержащую аминокислотную последовательность SEQ ID NO: 535, и (vi) CDR-L3, содержащую аминокислотную последовательность SEQ ID NO: 536. В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD30, содержит вариабельную область тяжелой цепи (VHCD30), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 537, и вариабельную область легкой цепи (VLCD30), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 538. В частности, антигенсвязывающий домен, способный к специфическому связыванию с CD30, содержит вариабельную область тяжелой цепи (VHCD30), содержащую аминокислотную последовательность SEQ ID NO: 537, и вариабельную область легкой цепи (VLCD30), содержащую аминокислотную последовательность SEQ ID NO: 538.
Нацеленные на TBPG (5T4) биспецифические агонистические CD28-антигенсвязывающие молекулы
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с опухолеассоциированным антигеном, представляет собой антигенсвязывающий домен, способный к специфическому связыванию с TBPG (5Т4).
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, в которой антигенсвязывающий домен, способный к специфическому связыванию с TBPG, содержит вариабельную область тяжелой цепи (VHTBPG), содержащую (i) CDR-Н1, содержащую аминокислотную последовательность SEQ ID NO: 539, (ii) CDR-H2, содержащую аминокислотную последовательность SEQ ID NO: 540, и (iii) CDR-H3, содержащую аминокислотную последовательность SEQ ID NO: 541, и вариабельную область легкой цепи (VLTBPG), содержащую (iv) CDR-L1, содержащую аминокислотную последовательность SEQ ID NO: 542, (v) CDR-L2, содержащую аминокислотную последовательность SEQ ID NO: 543, и (vi) CDR-L3, содержащую аминокислотную последовательность SEQ ID NO: 544. В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с TBPG, содержит вариабельную область тяжелой цепи (VHTBPG), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 545, и вариабельную область легкой цепи (VLTBPG), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 546. В частности, антигенсвязывающий домен, способный к специфическому связыванию с TBPG, содержит вариабельную область тяжелой цепи (VHTBPG), содержащую аминокислотную последовательность SEQ ID NO: 545, и вариабельную область легкой цепи (VLTBPG), содержащую аминокислотную последовательность SEQ ID NO: 546.
Нацеленные на ММ биспецифические агонистические CD28-антигенсвязывающие молекулы
В изобретении также предложены биспецифические агонистические CD28-антигенсвязывающие молекулы, которые являются исключительно полезными при лечении множественной миеломы. Эти молекулы содержат по меньшей мере один антигенсвязывающий домен, способный к специфическому связыванию с антигеном клеточной поверхности множественной миеломы (ММ), который приводит к перекрестному связыванию в присутствии экспрессирующих антиген клеточной поверхности ММ клеток, и Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию (Fc-сайленсинг). Таким образом, предотвращается неспецифическое перекрестное связывание посредством Fc-рецепторов и обеспечивается специфическая активация Т-клеток в присутствии экспрессирующих антиген клеточной поверхности ММ клеток.
Таким образом, в данном документе предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, содержащая антигенсвязывающий домен, способный к специфическому связыванию с CD28, антигенсвязывающий домен, способный к специфическому связыванию с антигеном клеточной поверхности множественной миеломы (ММ), и Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию. В одном аспекте описанная в данном документе биспецифическая агонистическая CD28-антигенсвязывающая молекула характеризуется одновалентным связыванием с CD28. В дополнительном аспекте описанная в данном документе биспецифическая агонистическая CD28-антигенсвязывающая молекула характеризуется одновалентным связыванием с антигеном клеточной поверхности множественной миеломы (ММ).
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула по определению ниже в данном документе, в которой Fc-домен принадлежит IgG, в частности представляет собой Fc-домен IgG1 или Fc-домен IgG4. В одном конкретном аспекте Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, представляет собой Fc-домен IgG1. Fc-домен содержит одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или снижает или устраняет эффекторную функцию. В одном аспекте Fc-домен содержит аминокислотные замены L234A и L235A (нумерация в соответствии с индексом EU по Kabat). В одном аспекте Fc-домен принадлежит подклассу IgG1 человека и содержит аминокислотные мутации L234A, L235A и P329G (нумерация в соответствии с индексом EU по Kabat).
В одном аспекте предложена определенная в данном документе биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антиген клеточной поверхности ММ выбран из группы, состоящей из CD38, ВСМА и GPRC5D.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с опухолеассоциированным антигеном, представляет собой антигенсвязывающий домен, способный к специфическому связыванию с CD38.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD38, содержит вариабельную область тяжелой цепи (VHCD38), содержащую (i) CDR-Н1, содержащую аминокислотную последовательность SEQ ID NO: 547, (ii) CDR-H2, содержащую аминокислотную последовательность SEQ ID NO: 548, и (iii) CDR-H3, содержащую аминокислотную последовательность SEQ ID NO: 549, и вариабельную область легкой цепи (VLCD38), содержащую (iv) CDR-L1, содержащую аминокислотную последовательность SEQ ID NO: 550, (v) CDR-L2, содержащую аминокислотную последовательность SEQ ID NO: 551, и (vi) CDR-L3, содержащую аминокислотную последовательность SEQ ID NO: 552. В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD38, содержит вариабельную область тяжелой цепи (VHCD38), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 553, и вариабельную область легкой цепи (VLCD38), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 554. В частности, антигенсвязывающий домен, способный к специфическому связыванию с CD38, содержит вариабельную область тяжелой цепи (VHCD38), содержащую аминокислотную последовательность SEQ ID NO: 553, и вариабельную область легкой цепи (VLCD38), содержащую аминокислотную последовательность SEQ ID NO: 554.
В еще одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с опухолеассоциированным антигеном, представляет собой антигенсвязывающий домен, способный к специфическому связыванию с ВСМА.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, в которой антигенсвязывающий домен, способный к специфическому связыванию с ВСМА, содержит вариабельную область тяжелой цепи (VHBCMA), содержащую (i) CDR-Н1, содержащую аминокислотную последовательность SEQ ID NO: 555, (ii) CDR-H2, содержащую аминокислотную последовательность SEQ ID NO: 556, и (iii) CDR-H3, содержащую аминокислотную последовательность SEQ ID NO: 557, и вариабельную область легкой цепи (VLBCMA), содержащую (iv) CDR-L1, содержащую аминокислотную последовательность SEQ ID NO: 558, (v) CDR-L2, содержащую аминокислотную последовательность SEQ ID NO: 559, и (vi) CDR-L3, содержащую аминокислотную последовательность SEQ ID NO: 560. В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с ВСМА, содержит вариабельную область тяжелой цепи (VHBCMA), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 561, и вариабельную область легкой цепи (VLBCMA), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 562. В частности, антигенсвязывающий домен, способный к специфическому связыванию с ВСМА, содержит вариабельную область тяжелой цепи (VHBCMA), содержащую аминокислотную последовательность SEQ ID NO: 561, и вариабельную область легкой цепи (VLBCMA), содержащую аминокислотную последовательность SEQ ID NO: 562.
Нацеленные на GPRC5D биспецифические агонистические CD28-антигенсвязывающие молекулы
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с опухолеассоциированным антигеном, представляет собой антигенсвязывающий домен, способный к специфическому связыванию с GPRC5D.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, в которой антигенсвязывающий домен, способный к специфическому связыванию с GPRC5D, содержит вариабельную область тяжелой цепи (VHGPRC5D), содержащую (i) CDR-H1, содержащую аминокислотную последовательность SEQ ID NO: 563, (ii) CDR-H2, содержащую аминокислотную последовательность SEQ ID NO: 564, и (iii) CDR-H3, содержащую аминокислотную последовательность SEQ ID NO: 565, и вариабельную область легкой цепи (VLGPRC5D), содержащую (iv) CDR-L1, содержащую аминокислотную последовательность SEQ ID NO: 566, (v) CDR-L2, содержащую аминокислотную последовательность SEQ ID NO: 567, и (vi) CDR-L3, содержащую аминокислотную последовательность SEQ ID NO: 568.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с GPRC5D, содержит вариабельную область тяжелой цепи (VHGPRC5D), содержащую аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 569, SEQ ID NO: 571, SEQ ID NO: 572 и SEQ ID NO: 573, и вариабельную область легкой цепи (VLGPRC5D), содержащую аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 570, SEQ ID NO: 574, SEQ ID NO: 575, SEQ ID NO: 576, SEQ ID NO: 577 и SEQ ID NO: 578.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с GPRC5D, содержит вариабельную область тяжелой цепи (VHGPRC5D), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 569, и вариабельную область легкой цепи (VLGPRC5D), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 570. В частности, антигенсвязывающий домен, способный к специфическому связыванию с GPRC5D, содержит вариабельную область тяжелой цепи (VHGPRC5D), содержащую аминокислотную последовательность SEQ ID NO: 569, и вариабельную область легкой цепи (VLGPRC5D), содержащую аминокислотную последовательность SEQ ID NO: 570.
В другом аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, в которой антигенсвязывающий домен, способный к специфическому связыванию с GPRC5D, содержит вариабельную область тяжелой цепи (VHGPRC5D), содержащую (i) CDR-H1, содержащую аминокислотную последовательность SEQ ID NO: 579, (ii) CDR-H2, содержащую аминокислотную последовательность SEQ ID NO: 580, и (iii) CDR-H3, содержащую аминокислотную последовательность SEQ ID NO: 581, и вариабельную область легкой цепи (VLGPRC5D), содержащую (iv) CDR-L1, содержащую аминокислотную последовательность SEQ ID NO: 582, (v) CDR-L2, содержащую аминокислотную последовательность SEQ ID NO: 583, и (vi) CDR-L3, содержащую аминокислотную последовательность SEQ ID NO: 584.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с GPRC5D, содержит вариабельную область тяжелой цепи (VHGPRC5D), содержащую аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 585, SEQ ID NO: 586, SEQ ID NO: 587, SEQ ID NO: 588, SEQ ID NO: 589 и SEQ ID NO: 590, и вариабельную область легкой цепи (VLGPRC5D), содержащую аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 591, SEQ ID NO: 592, SEQ ID NO: 593, SEQ ID NO: 594 и SEQ ID NO: 595.
В другом аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с GPRC5D, содержит
(а) вариабельную область тяжелой цепи (VHGPRC5D), содержащую аминокислотную последовательность SEQ ID NO: 569, и вариабельную область легкой цепи (VLGPRC5D), содержащую аминокислотную последовательность SEQ ID NO: 570, или
(б) вариабельную область тяжелой цепи (VHGPRC5D), содержащую аминокислотную последовательность SEQ ID NO: 573, и вариабельную область легкой цепи (VLGPRC5D), содержащую аминокислотную последовательность SEQ ID NO: 576, или
(в) вариабельную область тяжелой цепи (VHGPRC5D), содержащую аминокислотную последовательность SEQ ID NO: 569, и вариабельную область легкой цепи (VLGPRC5D), содержащую аминокислотную последовательность SEQ ID NO: 572, или
(г) вариабельную область тяжелой цепи (VHGPRC5D), содержащую аминокислотную последовательность SEQ ID NO: 586, и вариабельную область легкой цепи (VLGPRC5D), содержащую аминокислотную последовательность SEQ ID NO: 593, или
(д) вариабельную область тяжелой цепи (VHGPRC5D), содержащую аминокислотную последовательность SEQ ID NO: 587, и вариабельную область легкой цепи (VLGPRC5D), содержащую аминокислотную последовательность SEQ ID NO: 592.
Биспецифические агонистические CD28-антигенсвязывающие молекулы, одновалентные в отношении связывания с CD28 и одновалентные в отношении связывания с опухолеассоциированным антигеном (формат 1+1)
В другом аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один Fab-фрагмент, способный к специфическому связыванию с CD28,
(б) один кросс-Fab-фрагмент, способный к специфическому связыванию с опухолеассоциированным антигеном, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один Fab-фрагмент, способный к специфическому связыванию с CD28,
(б) один кросс-Fab-фрагмент, способный к специфическому связыванию с СЕА, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном конкретном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, содержащая первую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 65, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 66, вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 87, и вторую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 88 (молекула М).
В другом аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один Fab-фрагмент, способный к специфическому связыванию с CD28,
(б) один кросс-Fab-фрагмент, способный к специфическому связыванию с FAP, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном конкретном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, содержащая первую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 65, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 66, вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 67, и вторую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 68 (молекула С).
В дополнительном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) первый Fab-фрагмент, способный к специфическому связыванию с CD28,
(б) второй Fab-фрагмент, способный к специфическому связыванию с опухолеассоциированным антигеном, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию,
причем первый Fab-фрагмент, способный к специфическому связыванию с CD28, слит на С-конце тяжелой цепи Fab с N-концом тяжелой цепи Fab второго Fab-фрагмента, способного к специфическому связыванию с опухолеассоциированным антигеном, который в свою очередь слит на своем С-конце с N-концом одной из субъединиц Fc-домена.
В одном конкретном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, содержащая первую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 77, вторую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 78, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 75, и вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 79 (молекула Н).
В дополнительном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) Fab-фрагмент, способный к специфическому связыванию с CD28,
(б) домены VH и VL, способные к специфическому связыванию с опухолеассоциированным антигеном, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию,
причем Fab-фрагмент, способный к специфическому связыванию с CD28, слит на своем С-конце с N-концом первой субъединицы Fc-домена, и при этом один из доменов VH и VL, способных к специфическому связыванию с опухолеассоциированным антигеном, слит посредством пептидного линкера с С-концом первой субъединицы Fc-домена, а другой из доменов VH и VL, способных к специфическому связыванию с опухолеассоциированным антигеном, слит посредством пептидного линкера с С-концом второй субъединицы Fc-домена.
В одном аспекте пептидный линкер содержит аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 146, SEQ ID NO: 147, SEQ ID NO: 151 и SEQ ID NO: 152. В частности, пептидный линкер содержит SEQ ID NO: 152.
В одном конкретном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, содержащая легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 62, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 72, и вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 80 (молекула I).
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один Fab-фрагмент, способный к специфическому связыванию с CD28, содержащий
(i) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 47, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 54,
(ii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 53, или
(iii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 42, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 27,
(б) один кросс-Fab-фрагмент, способный к специфическому связыванию с опухолеассоциированным антигеном, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один Fab-фрагмент, способный к специфическому связыванию с CD28, содержащий
(i) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 47, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 54, или
(ii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 53, или
(iii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 42, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 27,
(б) один кросс-Fab-фрагмент, способный к специфическому связыванию с СЕА, содержащий
(i) вариабельную область тяжелой цепи (VHCEA), содержащую аминокислотную последовательность SEQ ID NO: 186, и вариабельную область легкой цепи (VLCEA), содержащую аминокислотную последовательность SEQ ID NO: 187, или
(ii) вариабельную область тяжелой цепи (VHCEA), содержащую аминокислотную последовательность SEQ ID NO: 200, и вариабельную область легкой цепи (VLCEA), содержащую аминокислотную последовательность SEQ ID NO: 201, или
(iii) вариабельную область тяжелой цепи (VHCEA), содержащую аминокислотную последовательность SEQ ID NO: 513, и вариабельную область легкой цепи (VLCEA), содержащую аминокислотную последовательность SEQ ID NO: 514,
и (в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, содержащая первую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 352, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 351, вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 353, и вторую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 354 (молекула 11А). В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, содержащая первую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 352, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 351, вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 355, и вторую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 356 (молекула 11В). В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, содержащая первую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 352, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 351, вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 357, и вторую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 358 (молекула 11С). В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, содержащая первую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 352, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 351, вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 359, и вторую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 354 (молекула 11D). В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, содержащая первую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 370, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 369, вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 353, и вторую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 356 (молекула 11I). В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, содержащая первую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 370, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 369, вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 359, и вторую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 354 (молекула 11J). В одном конкретном аспекте в изобретении предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, содержащая первую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 370, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 369, вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 357, и вторую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 358 (молекула 11K). В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, содержащая первую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 370, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 369, вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 359, и вторую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 356 (молекула 11L). В одном аспекте биспецифическая агонистическая CD28-антигенсвязывающая молекула содержит первую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 376, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 375, вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 357, и вторую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 358 (молекула 11R). В одном аспекте биспецифическая агонистическая CD28-антигенсвязывающая молекула содержит первую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 376, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 375, вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 355, и вторую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 356 (молекула 11S). В одном аспекте биспецифическая агонистическая CD28-антигенсвязывающая молекула содержит первую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 376, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 375, вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 355, и вторую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 354 (молекула 11Т).
В одном конкретном аспекте биспецифическая агонистическая CD28-антигенсвязывающая молекула содержит первую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 376, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 375, вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 355, и вторую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 356 (молекула 11S). В другом конкретном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, содержащая первую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 352, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 351, вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 355, и вторую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 356 (молекула 11В).
В другом аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один кросс-Fab-фрагмент, способный к специфическому связыванию с CD28,
(б) один Fab-фрагмент, способный к специфическому связыванию с опухолеассоциированным антигеном, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один кросс-Fab-фрагмент, способный к специфическому связыванию с CD28,
(б) один Fab-фрагмент, способный к специфическому связыванию с CEA, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один кросс-Fab-фрагмент, способный к специфическому связыванию с CD28, содержащий
(i) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 47, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 54, или
(ii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 53, или
(iii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 42, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 27,
(б) один Fab-фрагмент, способный к специфическому связыванию с СЕА, содержащий
(i) вариабельную область тяжелой цепи (VHCEA), содержащую аминокислотную последовательность SEQ ID NO: 186, и вариабельную область легкой цепи (VLCEA), содержащую аминокислотную последовательность SEQ ID NO: 187, или
(ii) вариабельную область тяжелой цепи (VHCEA), содержащую аминокислотную последовательность SEQ ID NO: 200, и вариабельную область легкой цепи (VLCEA), содержащую аминокислотную последовательность SEQ ID NO: 201, или
(iii) вариабельную область тяжелой цепи (VHCEA), содержащую аминокислотную последовательность SEQ ID NO: 513, и вариабельную область легкой цепи (VLCEA), содержащую аминокислотную последовательность SEQ ID NO: 514,
и (в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном аспекте биспецифическая агонистическая CD28-антигенсвязывающая молекула содержит первую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 361, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 360, вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 362, и вторую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 363 (молекула 11Е). В одном аспекте биспецифическая агонистическая CD28-антигенсвязывающая молекула содержит первую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 361, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 360, вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 364, и вторую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 365 (молекула 11F). В одном аспекте биспецифическая агонистическая CD28-антигенсвязывающая молекула содержит первую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 361, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 360, вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 366, и вторую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 367 (молекула 11G). В одном аспекте биспецифическая агонистическая CD28-антигенсвязывающая молекула содержит первую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 361, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 360, вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 368, и вторую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 363 (молекула 11Н). В одном аспекте биспецифическая агонистическая CD28-антигенсвязывающая молекула содержит первую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 372, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 371, вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 368, и вторую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 363 (молекула 11М). В одном аспекте биспецифическая агонистическая CD28-антигенсвязывающая молекула содержит первую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 372, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 371, вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 366, и вторую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 367 (молекула 11N). В одном аспекте биспецифическая агонистическая CD28-антигенсвязывающая молекула содержит первую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 372, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 371, вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 364, и вторую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 365 (молекула 11О).
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один Fab-фрагмент, способный к специфическому связыванию с CD28,
(б) один кросс-Fab-фрагмент, способный к специфическому связыванию с ЕрСАМ, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один Fab-фрагмент, способный к специфическому связыванию с CD28, содержащий
(i) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 47, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 54, или
(ii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 53, или
(iii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 42, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 27,
(б) один кросс-Fab-фрагмент, способный к специфическому связыванию с ЕрСАМ, содержащий вариабельную область тяжелой цепи (VHEpCAM), содержащую аминокислотную последовательность SEQ ID NO: 521, и вариабельную область легкой цепи (VLEpCAM), содержащую аминокислотную последовательность SEQ ID NO: 522,
и (в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один кросс-Fab-фрагмент, способный к специфическому связыванию с CD28,
(б) один Fab-фрагмент, способный к специфическому связыванию с ЕрСАМ, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один кросс-Fab-фрагмент, способный к специфическому связыванию с CD28, содержащий
(i) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 47, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 54, или
(ii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 53, или
(iii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 42, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 27,
(б) один Fab-фрагмент, способный к специфическому связыванию с ЕрСАМ, содержащий вариабельную область тяжелой цепи (VHEpCAM), содержащую аминокислотную последовательность SEQ ID NO: 521, и вариабельную область легкой цепи (VLEpCAM), содержащую аминокислотную последовательность SEQ ID NO: 522,
и (в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном аспекте биспецифическая агонистическая CD28-антигенсвязывающая молекула содержит первую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 367, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 366, вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 390, и вторую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 391 (молекула 14А).
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один Fab-фрагмент, способный к специфическому связыванию с CD28,
(б) один кросс-Fab-фрагмент, способный к специфическому связыванию с HER3, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один Fab-фрагмент, способный к специфическому связыванию с CD28, содержащий
(i) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 47, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 54, или
(ii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 53, или
(iii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 42, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 27,
(б) один кросс-Fab-фрагмент, способный к специфическому связыванию с HER3, содержащий вариабельную область тяжелой цепи (VHHER3), содержащую аминокислотную последовательность SEQ ID NO: 529, и вариабельную область легкой цепи (VLHER3), содержащую аминокислотную последовательность SEQ ID NO: 530,
и (в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном аспекте биспецифическая агонистическая CD28-антигенсвязывающая молекула содержит первую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 357, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 358, вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 392, и вторую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 393 (молекула 14В).
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один кросс-Fab-фрагмент, способный к специфическому связыванию с CD28,
(б) один Fab-фрагмент, способный к специфическому связыванию с HER3, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один кросс-Fab-фрагмент, способный к специфическому связыванию с CD28, содержащий
(i) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 47, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 54, или
(ii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 53, или
(iii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 42, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 27,
(б) один Fab-фрагмент, способный к специфическому связыванию с HER3, содержащий вариабельную область тяжелой цепи (VHHER3), содержащую аминокислотную последовательность SEQ ID NO: 529, и вариабельную область легкой цепи (VLHER3), содержащую аминокислотную последовательность SEQ ID NO: 530,
и (в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один Fab-фрагмент, способный к специфическому связыванию с CD28,
(б) один кросс-Fab-фрагмент, способный к специфическому связыванию с CD30, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один Fab-фрагмент, способный к специфическому связыванию с CD28, содержащий
(i) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 47, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 54, или
(ii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 53, или
(iii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 42, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 27,
(б) один кросс-Fab-фрагмент, способный к специфическому связыванию с CD30, содержащий вариабельную область тяжелой цепи (VHCD30), содержащую аминокислотную последовательность SEQ ID NO: 537, и вариабельную область легкой цепи (VLCD30), содержащую аминокислотную последовательность SEQ ID NO: 538,
и (в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном аспекте биспецифическая агонистическая CD28-антигенсвязывающая молекула содержит первую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 357, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 358, вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 394, и вторую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 395 (молекула 14С).
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один кросс-Fab-фрагмент, способный к специфическому связыванию с CD28,
(б) один Fab-фрагмент, способный к специфическому связыванию с CD30, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один кросс-Fab-фрагмент, способный к специфическому связыванию с CD28, содержащий
(i) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 47, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 54, или
(ii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 53, или
(iii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 42, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 27,
(б) один Fab-фрагмент, способный к специфическому связыванию с CD30, содержащий вариабельную область тяжелой цепи (VHCD30), содержащую аминокислотную последовательность SEQ ID NO: 537, и вариабельную область легкой цепи (VLCD30), содержащую аминокислотную последовательность SEQ ID NO: 538,
и (в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один Fab-фрагмент, способный к специфическому связыванию с CD28,
(б) один кросс-Fab-фрагмент, способный к специфическому связыванию с TPBG, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один Fab-фрагмент, способный к специфическому связыванию с CD28, содержащий
(i) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 47, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 54, или
(ii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 53, или
(iii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 42, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 27,
(б) один кросс-Fab-фрагмент, способный к специфическому связыванию с TPBG, содержащий вариабельную область тяжелой цепи (VHTPBG), содержащую аминокислотную последовательность SEQ ID NO: 545, и вариабельную область легкой цепи (VLTPBG), содержащую аминокислотную последовательность SEQ ID NO: 546,
и (в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном аспекте биспецифическая агонистическая CD28-антигенсвязывающая молекула содержит первую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 357, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 358, вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 396, и вторую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 397 (молекула 14D).
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один кросс-Fab-фрагмент, способный к специфическому связыванию с CD28,
(б) один Fab-фрагмент, способный к специфическому связыванию с TPBG, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один кросс-Fab-фрагмент, способный к специфическому связыванию с CD28, содержащий
(i) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 47, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 54, или
(ii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 53, или
(iii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 42, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 27,
(б) один Fab-фрагмент, способный к специфическому связыванию с TPBG, содержащий вариабельную область тяжелой цепи (VHTPBG), содержащую аминокислотную последовательность SEQ ID NO: 545, и вариабельную область легкой цепи (VLTPBG), содержащую аминокислотную последовательность SEQ ID NO: 546,
и (в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один Fab-фрагмент, способный к специфическому связыванию с CD28,
(б) один кросс-Fab-фрагмент, способный к специфическому связыванию с CD38, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один Fab-фрагмент, способный к специфическому связыванию с CD28, содержащий
(i) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 47, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 54, или
(ii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 53, или
(iii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 42, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 27,
(б) один кросс-Fab-фрагмент, способный к специфическому связыванию с CD38, содержащий вариабельную область тяжелой цепи (VHCD38), содержащую аминокислотную последовательность SEQ ID NO: 553, и вариабельную область легкой цепи (VLCD38), содержащую аминокислотную последовательность SEQ ID NO: 554,
и (в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном аспекте биспецифическая агонистическая CD28-антигенсвязывающая молекула содержит первую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 357, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 358, вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 400, и вторую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 401 (молекула 16С).
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один кросс-Fab-фрагмент, способный к специфическому связыванию с CD28,
(б) один Fab-фрагмент, способный к специфическому связыванию с CD38, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один кросс-Fab-фрагмент, способный к специфическому связыванию с CD28, содержащий
(i) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 47, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 54, или
(ii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 53, или
(iii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 42, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 27,
(б) один Fab-фрагмент, способный к специфическому связыванию с CD38, содержащий вариабельную область тяжелой цепи (VHCD38), содержащую аминокислотную последовательность SEQ ID NO: 553, и вариабельную область легкой цепи (VLCD38), содержащую аминокислотную последовательность SEQ ID NO: 554,
и (в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один Fab-фрагмент, способный к специфическому связыванию с CD28,
(б) один кросс-Fab-фрагмент, способный к специфическому связыванию с ВСМА, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один Fab-фрагмент, способный к специфическому связыванию с CD28, содержащий
(i) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 47, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 54, или
(ii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 53, или
(iii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 42, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 27,
(б) один кросс-Fab-фрагмент, способный к специфическому связыванию с ВСМА, содержащий вариабельную область тяжелой цепи (VHBCMA), содержащую аминокислотную последовательность SEQ ID NO: 561, и вариабельную область легкой цепи (VLBCMA), содержащую аминокислотную последовательность SEQ ID NO: 562,
и (в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один кросс-Fab-фрагмент, способный к специфическому связыванию с CD28,
(б) один Fab-фрагмент, способный к специфическому связыванию с ВСМА, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один кросс-Fab-фрагмент, способный к специфическому связыванию с CD28, содержащий
(i) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 47, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 54, или
(ii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 53, или
(iii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 42, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 27,
(б) один Fab-фрагмент, способный к специфическому связыванию с ВСМА, содержащий вариабельную область тяжелой цепи (VHBCMA), содержащую аминокислотную последовательность SEQ ID NO: 561, и вариабельную область легкой цепи (VLBCMA), содержащую аминокислотную последовательность SEQ ID NO: 562,
и (в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном аспекте биспецифическая агонистическая CD28-антигенсвязывающая молекула содержит первую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 367, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 366, вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 402, и вторую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 403 (молекула 16D).
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один Fab-фрагмент, способный к специфическому связыванию с CD28,
(б) один кросс-Fab-фрагмент, способный к специфическому связыванию с GPRC5D, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один Fab-фрагмент, способный к специфическому связыванию с CD28, содержащий
(i) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 47, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 54, или
(ii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 53, или
(iii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 42, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 27,
(б) один кросс-Fab-фрагмент, способный к специфическому связыванию с GPRC5D, содержащий вариабельную область тяжелой цепи (VHGPRC5D), содержащую аминокислотную последовательность SEQ ID NO: 569, и вариабельную область легкой цепи (VLGPRC5D), содержащую аминокислотную последовательность SEQ ID NO: 570,
и (в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном аспекте биспецифическая агонистическая CD28-антигенсвязывающая молекула содержит первую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 365, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 364, вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 398, и вторую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 399 (молекула 16В).
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один кросс-Fab-фрагмент, способный к специфическому связыванию с CD28,
(б) один Fab-фрагмент, способный к специфическому связыванию с GPRC5D, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один кросс-Fab-фрагмент, способный к специфическому связыванию с CD28, содержащий
(i) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 47, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 54, или
(ii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 53, или
(iii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 42, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 27,
(б) один Fab-фрагмент, способный к специфическому связыванию с GPRC5D, содержащий вариабельную область тяжелой цепи (VHGPRC5D), содержащую аминокислотную последовательность SEQ ID NO: 569, и вариабельную область легкой цепи (VLGPRC5D), содержащую аминокислотную последовательность SEQ ID NO: 570,
и (в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном аспекте биспецифическая агонистическая CD28-антигенсвязывающая молекула содержит первую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 367, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 366, вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 398, и вторую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 399 (молекула 16А).
Биспецифические агонистические CD28-антигенсвязывающие молекулы, двухвалентные в отношении связывания с CD28 и одновалентные в отношении связывания с опухолеассоциированным антигеном (формат 1+2)
В другом аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) первый Fab-фрагмент, способный к специфическому связыванию с CD28,
(б) второй и третий Fab-фрагменты, способные к специфическому связыванию с опухолеассоциированным антигеном, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию,
причем первый Fab-фрагмент, способный к специфическому связыванию с CD28, слит на С-конце тяжелой цепи Fab с N-концом тяжелой цепи Fab второго Fab-фрагмента, способного к специфическому связыванию с опухолеассоциированным антигеном, который в свою очередь слит на своем С-конце с N-концом первой субъединицы Fc-домена, и третий Fab-фрагмент, способный к специфическому связыванию с опухолеассоциированным антигеном, слит на С-конце тяжелой цепи Fab с N-концом второй субъединицы Fc-домена.
В одном конкретном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, содержащая две легкие цепи, каждая из которых содержит аминокислотную последовательность SEQ ID NO: 78, одну легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 77, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 75, и вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 76 (молекула G).
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) первый кросс-Fab-фрагмент, способный к специфическому связыванию с CD28, содержащий
(i) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 47, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 54, или
(ii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 53, или
(iii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 42, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 27,
(б) второй и третий Fab-фрагменты, способные к специфическому связыванию с СЕА, содержащие
(i) вариабельную область тяжелой цепи (VHCEA), содержащую аминокислотную последовательность SEQ ID NO: 186, и вариабельную область легкой цепи (VLCEA), содержащую аминокислотную последовательность SEQ ID NO: 187, или
(ii) вариабельную область тяжелой цепи (VHCEA), содержащую аминокислотную последовательность SEQ ID NO: 200, и вариабельную область легкой цепи (VLCEA), содержащую аминокислотную последовательность SEQ ID NO: 201, или
(iii) вариабельную область тяжелой цепи (VHCEA), содержащую аминокислотную последовательность SEQ ID NO: 513, и вариабельную область легкой цепи (VLCEA), содержащую аминокислотную последовательность SEQ ID NO: 514,
и (в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном конкретном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, содержащая две легкие цепи, каждая из которых содержит аминокислотную последовательность SEQ ID NO: 361, одну легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 368, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 362, и вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 373 (молекула 11P).
В одном конкретном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, содержащая две легкие цепи, каждая из которых содержит аминокислотную последовательность SEQ ID NO: 361, одну легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 368, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 360, и вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 374 (молекула 11Q).
Нацеленные на FAP и СЕА агонистические CD28-антигенсвязывающие молекулы
В данном документе также предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула с одновалентным связыванием с CD28, содержащая
(а) один антигенсвязывающий домен, способный к специфическому связыванию с CD28,
(б) один антигенсвязывающий домен, способный к специфическому связыванию с первым опухолеассоциированным антигеном, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию, характеризующийся тем, что он дополнительно содержит один антигенсвязывающий домен, способный к специфическому связыванию со вторым опухолеассоциированным антигеном.
В одном конкретном аспекте предложена триспецифическая агонистическая CD28-антигенсвязывающая молекула с одновалентным связыванием с CD28, содержащая
(а) один антигенсвязывающий домен, способный к специфическому связыванию с CD28,
(б) один антигенсвязывающий домен, способный к специфическому связыванию с СЕА, и один антигенсвязывающий домен, способный к специфическому связыванию с FAP,
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном конкретном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, содержащая первую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 88, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 87, вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 388, и вторую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 389 (молекула Y).
Нацеленные на поверхностный антиген В-клеток биспецифические агонистические CD28-антигенсвязывающие молекулы
В изобретении предложены новые биспецифические агонистические CD28-антигенсвязывающие молекулы с исключительно преимущественными свойствами, такими как технологичность, стабильность, аффинность связывания, биологическая активность, эффективность нацеливания, сниженная токсичность, расширенный диапазон дозировок, которые можно вводить пациенту, и, следовательно, возможно повышенная эффективность. Новые биспецифические агонистические CD28-антигенсвязывающие молекулы содержат Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию (Fc-сайленсинг) и, таким образом, предотвращается перекрестное связывание посредством Fc-рецепторов. Вместо этого они содержат по меньшей мере один антигенсвязывающий домен, способный к специфическому связыванию с поверхностным антигеном В-клеток, таким как CD19 или CD79b, который приводит к перекрестному связыванию в присутствии CD19- или CD79b-экспрессирующих В-клеток. Таким образом достигается специфическая активация Т-клеток в присутствии CD19- или CD79b-экспрессирующих В-клеток.
В данном документе предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, содержащая антигенсвязывающий домен, способный к специфическому связыванию с CD28, антигенсвязывающий домен, способный к специфическому связыванию с поверхностным антигеном В-клеток, и Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию. В одном аспекте описанная в данном документе биспецифическая агонистическая CD28-антигенсвязывающая молекула характеризуется одновалентным связыванием с CD28. В дополнительном аспекте описанная в данном документе биспецифическая агонистическая CD28-антигенсвязывающая молекула характеризуется одновалентным связыванием с поверхностным антигеном В-клеток.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула по определению ниже в данном документе, в которой Fc-домен принадлежит IgG, в частности представляет собой Fc-домен IgG1 или Fc-домен IgG4. В одном конкретном аспекте Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, представляет собой Fc-домен IgG1. Fc-домен содержит одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или снижает или устраняет эффекторную функцию. В одном аспекте Fc-домен содержит аминокислотные замены L234A и L235A (нумерация в соответствии с индексом EU по Kabat). В одном аспекте Fc-домен принадлежит подклассу IgG1 человека и содержит аминокислотные мутации L234A, L235A и P329G (нумерация в соответствии с индексом EU по Kabat).
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула по определению выше в данном документе, в которой по меньшей мере один антигенсвязывающий домен, способный к специфическому связыванию с CD28, содержит
(i) вариабельную область тяжелой цепи (VHCD28), содержащую определяющую комплементарность область тяжелой цепи CDR-H1 с SEQ ID NO: 20, CDR-H2 с SEQ ID NO: 21 и CDR-H3 с SEQ ID NO: 22, и вариабельную область легкой цепи (VLCD28), содержащую определяющую комплементарность область легкой цепи CDR-L1 с SEQ ID NO: 23, CDR-L2 с SEQ ID NO: 24 и CDR-L3 с SEQ ID NO: 25; или
(ii) вариабельную область тяжелой цепи (VHCD28), содержащую CDR-H1 с SEQ ID NO: 36, CDR-H2 с SEQ ID NO: 37 и CDR-H3 с SEQ ID NO: 38, и вариабельную область легкой цепи (VLCD28), содержащую CDR-L1 с SEQ ID NO: 39, CDR-L2 с SEQ ID NO: 40 и CDR-L3 с SEQ ID NO: 41.
В одном аспекте антигенсвязывающий домен, способный к специфическому связыванию с CD28, биспецифической агонистической CD28-антигенсвязывающей молекулы содержит вариабельную область тяжелой цепи (VHCD28), содержащую CDR-H1 с SEQ ID NO: 20, CDR-H2 с SEQ ID NO: 21 и CDR-H3 с SEQ ID NO: 22, и вариабельную область легкой цепи (VLCD28), содержащую CDR-L1 с SEQ ID NO: 23, CDR-L2 с SEQ ID NO: 24 и CDR-L3 с SEQ ID NO: 25. В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула по определению выше в данном документе, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD28, содержит вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 26, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 27. В одном аспекте антигенсвязывающий домен, способный к специфическому связыванию с CD28, содержит вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 26, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 27.
В другом аспекте антигенсвязывающий домен, способный к специфическому связыванию с CD28, биспецифической агонистической CD28-антигенсвязывающей молекулы содержит вариабельную область тяжелой цепи (VHCD28), содержащую CDR-H1 с SEQ ID NO: 28, CDR-H2 с SEQ ID NO: 29 и CDR-H3 с SEQ ID NO: 30, и вариабельную область легкой цепи (VLCD28), содержащую CDR-L1 с SEQ ID NO: 31, CDR-L2 с SEQ ID NO: 32 и CDR-L3 с SEQ ID NO: 33. В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула по определению выше в данном документе, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD28, содержит вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 34, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 35. В одном аспекте антигенсвязывающий домен, способный к специфическому связыванию с CD28, содержит вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 34, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 35.
В одном аспекте антигенсвязывающий домен, способный к специфическому связыванию с CD28, биспецифической агонистической CD28-антигенсвязывающей молекулы содержит вариабельную область тяжелой цепи (VHCD28), содержащую CDR-H1 с SEQ ID NO: 36, CDR-H2 с SEQ ID NO: 37 и CDR-H3 с SEQ ID NO: 38, и вариабельную область легкой цепи (VLCD28), содержащую CDR-L1 с SEQ ID NO: 39, CDR-L2 с SEQ ID NO: 40 и CDR-L3 с SEQ ID NO: 41.
В дополнительном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD28, содержит вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 42, SEQ ID NO: 43, SEQ ID NO: 44, SEQ ID NO: 45, SEQ ID NO: 46, SEQ ID NO: 47, SEQ ID NO: 48, SEQ ID NO: 49, SEQ ID NO: 50 и SEQ ID NO: 51, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 27, SEQ ID NO: 52, SEQ ID NO: 53, SEQ ID NO: 54, SEQ ID NO: 55, SEQ ID NO: 56, SEQ ID NO: 57, SEQ ID NO: 58, SEQ ID NO: 59, SEQ ID NO: 60 и SEQ ID NO: 61.
В другом аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD28, содержит
(а) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 47, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 54, или
(б) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 47, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 27, или
(в) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 51, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 61, или
(г) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 53, или
(д) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 54, или
(е) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 59, или
(ж) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 27, или
(и) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 43, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 27, или
(к) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 42, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 53, или
(л) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 42, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 59, или
(м) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 42, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 27.
В одном аспекте биспецифическая агонистическая CD28-антигенсвязывающая молекула содержит антигенсвязывающий домен, способный к специфическому связыванию с CD28, содержащий вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 53, или вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 47, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 54, или вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 47, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 27, или вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 42, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 27. В одном конкретном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD28, содержит вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 53. В другом конкретном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD28, содержит вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 47, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 54. В дополнительном конкретном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD28, содержит вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 47, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 27. В другом конкретном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD28, содержит вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 42, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 27.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с поверхностным антигеном В-клеток, представляет собой антигенсвязывающий домен, способный к специфическому связыванию с CD19.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD 19, содержит (а) вариабельную область тяжелой цепи (VHCD19), содержащую (i) CDR-H1, содержащую аминокислотную последовательность SEQ ID NO: 406, (ii) CDR-H2, содержащую аминокислотную последовательность SEQ ID NO: 407, и (iii) CDR-H3, содержащую аминокислотную последовательность SEQ ID NO: 408, и вариабельную область легкой цепи (VLCD19), содержащую (iv) CDR-L1, содержащую аминокислотную последовательность SEQ ID NO: 409, (v) CDR-L2, содержащую аминокислотную последовательность SEQ ID NO: 410, и (vi) CDR-L3, содержащую аминокислотную последовательность SEQ ID NO: 411, или (б) вариабельную область тяжелой цепи (VHCD19), содержащую (i) CDR-H1, содержащую аминокислотную последовательность SEQ ID NO: 414, (ii) CDR-H2, содержащую аминокислотную последовательность SEQ ID NO: 415, и (iii) CDR-НЗ, содержащую аминокислотную последовательность SEQ ID NO: 416, и вариабельную область легкой цепи (VLCD19), содержащую (iv) CDR-L1, содержащую аминокислотную последовательность SEQ ID NO: 417, (v) CDR-L2, содержащую аминокислотную последовательность SEQ ID NO: 418, и (vi) CDR-L3, содержащую аминокислотную последовательность SEQ ID NO: 419. В частности, антигенсвязывающий домен, способный к специфическому связыванию с CD 19, содержит (а) вариабельную область тяжелой цепи (VHCD19), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 98% или 100% идентичной аминокислотной последовательности SEQ ID NO: 412, и вариабельную область легкой цепи (VLCD19), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 98% или 100% идентичной аминокислотной последовательности SEQ ID NO: 413, или (б) вариабельную область тяжелой цепи (VHCD19), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 98% или 100% идентичной аминокислотной последовательности SEQ ID NO: 420, и вариабельную область легкой цепи (VLCD19), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 98% или 100% идентичной аминокислотной последовательности SEQ ID NO: 421. В одном конкретном аспекте антигенсвязывающий домен, способный к специфическому связыванию с CD19, содержит вариабельную область тяжелой цепи (VHCD19), содержащую аминокислотную последовательность SEQ ID NO: 412, и вариабельную область легкой цепи (VLCD19), содержащую аминокислотную последовательность SEQ ID NO: 413.
В другом аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, в которой антигенсвязывающий домен, способный к специфическому связыванию с поверхностным антигеном В-клеток, представляет собой антигенсвязывающий домен, способный к специфическому связыванию с CD79b.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD79b, содержит вариабельную область тяжелой цепи (VHCD79b), содержащую (i) CDR-Н1, содержащую аминокислотную последовательность SEQ ID NO: 422, (ii) CDR-H2, содержащую аминокислотную последовательность SEQ ID NO: 423, и (iii) CDR-H3, содержащую аминокислотную последовательность SEQ ID NO: 424, и вариабельную область легкой цепи (VLCD79b), содержащую (iv) CDR-L1, содержащую аминокислотную последовательность SEQ ID NO: 425, (v) CDR-L2, содержащую аминокислотную последовательность SEQ ID NO: 426, и (vi) CDR-L3, содержащую аминокислотную последовательность SEQ ID NO: 427. В частности, антигенсвязывающий домен, способный к специфическому связыванию с CD79b, содержит вариабельную область тяжелой цепи (VHCD79b), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 98% или 100% идентичной аминокислотной последовательности SEQ ID NO: 428, и вариабельную область легкой цепи (VLCD79b), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 98% или 100% идентичной аминокислотной последовательности SEQ ID NO: 429. В одном аспекте антигенсвязывающий домен, способный к специфическому связыванию с CD79b, содержит вариабельную область тяжелой цепи (VHCD79b), содержащую аминокислотную последовательность SEQ ID NO: 428, и вариабельную область легкой цепи (VLCD79b), содержащую аминокислотную последовательность SEQ ID NO: 429.
В дополнительном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула по определению выше в данном документе, в которой по меньшей мере один антигенсвязывающий домен, способный к специфическому связыванию с CD28, представляет собой Fab-фрагмент или Kpocc-Fab-фрагмент. В одном конкретном аспекте антигенсвязывающий домен, способный к специфическому связыванию с CD28, представляет собой Fab-фрагмент, а антигенсвязывающий домен, способный к специфическому связыванию с поверхностным антигеном В-клеток, представляет собой Kpocc-Fab-фрагмент.
Биспецифические агонистические CD28-антигенсвязывающие молекулы, одновалентные в отношении связывания с CD28 и одновалентные в отношении связывания с поверхностным антигеном В-клеток (формат 1+1)
В другом аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один Fab-фрагмент, способный к специфическому связыванию с CD28,
(б) один Kpocc-Fab-фрагмент, способный к специфическому связыванию с поверхностным антигеном В-клеток, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один Fab-фрагмент, способный к специфическому связыванию с CD28,
(б) один Kpocc-Fab-фрагмент, способный к специфическому связыванию с CD19, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один Fab-фрагмент, способный к специфическому связыванию с CD28, содержащий
(i) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 47, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 54, или
(ii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 53, или
(iii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 42, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 27,
(б) один Kpocc-Fab-фрагмент, способный к специфическому связыванию с CD19, содержащий
(i) вариабельную область тяжелой цепи (VHCD19), содержащую аминокислотную последовательность SEQ ID NO: 412, и вариабельную область легкой цепи (VLCD19), содержащую аминокислотную последовательность SEQ ID NO: 413, или
(ii) вариабельную область тяжелой цепи (VHCD19), содержащую аминокислотную последовательность SEQ ID NO: 420, и вариабельную область легкой цепи (VLCD19), содержащую аминокислотную последовательность SEQ ID NO: 421,
и (в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном аспекте в изобретении предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, содержащая первую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 65, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 118, вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 430, и вторую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 431 (молекула 18А).
В одном конкретном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, содержащая первую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 121, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 116, вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 430, и вторую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 431 (молекула 18В).
В другом конкретном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, содержащая первую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 122, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 114, вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 430, и вторую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 431 (молекула 18С).
В одном дополнительном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, содержащая первую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 65, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 114, вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 430, и вторую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 431 (молекула 18D).
В другом аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, содержащая первую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 123, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 118, вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 430, и вторую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 431 (молекула 18Е).
В другом аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один Kpocc-Fab-фрагмент, способный к специфическому связыванию с CD28,
(б) один Fab-фрагмент, способный к специфическому связыванию с CD 19, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один Kpocc-Fab-фрагмент, способный к специфическому связыванию с CD28, содержащий
(i) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 47, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 54, или
(ii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 53, или
(iii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 42, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 27,
(б) один Fab-фрагмент, способный к специфическому связыванию с CD 19, содержащий
(i) вариабельную область тяжелой цепи (VHCD19), содержащую аминокислотную последовательность SEQ ID NO: 412, и вариабельную область легкой цепи (VLCD19), содержащую аминокислотную последовательность SEQ ID NO: 413, или
(ii) вариабельную область тяжелой цепи (VHCD19), содержащую аминокислотную последовательность SEQ ID NO: 420, и вариабельную область легкой цепи (VLCD19), содержащую аминокислотную последовательность SEQ ID NO: 421,
и (в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один Fab-фрагмент, способный к специфическому связыванию с CD28,
(б) один Kpocc-Fab-фрагмент, способный к специфическому связыванию с CD79b, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один Fab-фрагмент, способный к специфическому связыванию с CD28, содержащий
(i) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 47, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 54, или
(ii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 53, или
(iii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 42, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 27,
(б) один Kpocc-Fab-фрагмент, способный к специфическому связыванию с CD79b, содержащий вариабельную область тяжелой цепи (VHCD79b), содержащую аминокислотную последовательность SEQ ID NO: 428, и вариабельную область легкой цепи (VLCD79b), содержащую аминокислотную последовательность SEQ ID NO: 429,
и (в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В другом конкретном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, содержащая первую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 121, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 116, вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 432, и вторую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 433 (молекула 18F).
В другом аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один Kpocc-Fab-фрагмент, способный к специфическому связыванию с CD28,
(б) один Fab-фрагмент, способный к специфическому связыванию с CD19, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) один Kpocc-Fab-фрагмент, способный к специфическому связыванию с CD28, содержащий
(i) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 47, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 54, или
(ii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 53, или
(iii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 42, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 27,
(б) один Fab-фрагмент, способный к специфическому связыванию с CD79b, содержащий вариабельную область тяжелой цепи (VHCD79b), содержащую аминокислотную последовательность SEQ ID NO: 428, и вариабельную область легкой цепи (VLCD79b), содержащую аминокислотную последовательность SEQ ID NO: 429,
и (в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
Биспецифические агонистические СВ28-антигенсвязывающие молекулы, одновалентные в отношении связывания с CD28 и двухвалентные в отношении связывания с поверхностным антигеном В-клеток (формат 1+2)
В одном аспекте биспецифическая агонистическая CD28-антигенсвязывающая молекула характеризуется двухвалентным связыванием с поверхностным антигеном В-клеток.
В другом аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, описанная в данном документе, содержащая
(а) первый Fab-фрагмент, способный к специфическому связыванию с CD28,
(б) второй и третий Fab-фрагменты, способные к специфическому связыванию с поверхностным антигеном В-клеток, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию,
причем первый Fab-фрагмент, способный к специфическому связыванию с CD28, слит на С-конце тяжелой цепи Fab с N-концом тяжелой цепи Fab второго Fab-фрагмента, способного к специфическому связыванию с опухолеассоциированным антигеном, который в свою очередь слит на своем С-конце с N-концом первой субъединицы Fc-домена, и третий Fab-фрагмент, способный к специфическому связыванию с опухолеассоциированным антигеном, слит на С-конце тяжелой цепи Fab с N-концом второй субъединицы Fc-домена.
Модификации Fc-домена, снижающие связывание Fc-рецептора и/или эффекторную функцию
Fc-домен биспецифической агонистической CD28-антигенсвязывающей молекулы по изобретению состоит из пары полипептидных цепей, содержащих домены тяжелой цепи молекулы иммуноглобулина. Например, Fc-домен молекулы иммуноглобулина G (IgG) представляет собой димер, каждая субъединица которого содержит константные домены СН2 и СН3 тяжелой цепи IgG. Две субъединицы Fc-домена способны к стабильной ассоциации друг с другом. Fc-домен придает благоприятные фармакокинетические свойства антигенсвязывающим молекулам по изобретению, включая длительное сывороточное время полужизни, которое способствует надлежащему накоплению в целевой ткани и благоприятному соотношению распределения в ткани и крови. Однако, с другой стороны, он может приводить к нежелательному нацеливанию биспецифических антител по изобретению на клетки, экспрессирующие Fc-рецепторы, вместо предпочтительных несущих антиген клеток.
Соответственно, Fc-домен биспецифической агонистической CD28-антигенсвязывающей молекулы по изобретению демонстрирует сниженную аффинность связывания с Fc-рецептором и/или сниженную эффекторную функцию по сравнению с Fc-доменом нативного IgG1. В одном аспекте Fc практически не связывается с Fc-рецептором и/или не индуцирует эффекторную функцию. В конкретном аспекте Fc-рецептор представляет собой Fcy-рецептор. В одном аспекте Fc-рецептор представляет собой человеческий Fc-рецептор. В конкретном аспекте Fc-рецептор представляет собой активирующий человеческий Fcγ-рецептор, в частности, человеческий FcγRIIIa, FcγRI или FcγRIIa, конкретно - человеческий FcγRIIIa. В одном аспекте Fc-домен не индуцирует эффекторную функцию. Сниженная эффекторная функция может включать, но не ограничивается этим, одно или более из следующего: сниженную комплементзависимую цитотоксичность (КЗЦ), сниженную антителозависимую клеточноопосредованную цитотоксичность (АЗКЦ), сниженный антителозависимый клеточный фагоцитоз (АЗКФ), сниженную секрецию цитокинов, сниженное опосредованное иммунным комплексом поглощение антигена антигенпрезентирующими клетками, сниженное связывание с NK-клетками, сниженное связывание с макрофагами, сниженное связывание с моноцитами, сниженное связывание с полиморфно-ядерными клетками, сниженную прямую сигнализацию, индуцирующую апоптоз, сниженное созревание дендритных клеток или сниженное примирование Т-клеток.
В определенных аспектах одну или более аминокислотных модификаций можно вносить в Fc-область антитела, предложенного в данном документе, с получением, таким образом, варианта по Fc-области. Вариант по Fc-области может содержать последовательность человеческой Fc-области (например, Fc-области человеческого IgG1, IgG2, IgG3 или IgG4), содержащую аминокислотную модификацию (например, замену) в одной или более аминокислотных позициях.
В одном конкретном аспекте в изобретении предложена антигенсвязывающая молекула, в которой Fc-область содержит одну или более аминокислотных замен, которые снижают связывание с Fc-рецептором, в частности с Fcγ-рецептором. В одном аспекте в изобретении предложено антитело, в котором Fc-область содержит одну или более аминокислотных замен и при этом АЗКЦ, индуцируемая антителом, снижена до 0-20% АЗКЦ, индуцируемой антителом, содержащим Fc-область человеческого IgG1 дикого типа.
В одном аспекте Fc-домен антигенсвязывающей молекулы по изобретению содержит одну или более аминокислотных мутаций, которые снижают аффинность связывания Fc-домена с Fc-рецептором и/или эффекторную функцию. Как правило, в каждой из двух субъединиц Fc-домена присутствуют одни и те же одна или более аминокислотных мутаций. Как правило, Fc-домен содержит аминокислотную замену в позиции Е233, L234, L235, N297, Р331 и Р329 (нумерация EU). В частности, Fc-домен содержит аминокислотные замены в позициях 234 и 235 (нумерация EU) и/или 329 (нумерация EU) тяжелых цепей IgG. конкретнее, предложена антигенсвязывающая молекула в соответствии с изобретением, которая содержит Fc-домен с аминокислотными заменами L234A, L235A и P329G («P329G LALA», нумерация EU) в тяжелых цепях IgG. Аминокислотные замены L234A и L235A относятся к так называемой мутации LALA. Комбинация аминокислотных замен «P329G LALA» практически полностью устраняет связывание Fcγ-рецептор а Fc-доменом человеческого IgG1 и описана в публикации Международной патентной заявки №WO 2012/130831 А1, в которой также описаны способы получения таких мутантных Fc-доменов и способы определения их свойств, таких как связывание Fc-рецептора или эффекторные функции.
Fc-домены со сниженными связыванием Fc-рецептора и/или эффекторной функцией также включают антитела с заменой одного или более остатков Fc-домена 238, 265, 269, 270, 297, 327 и 329 (патент США №6737056). Такие Fc-мутанты включают Fc-мутантов с заменами в двух или более аминокислотных позициях 265, 269, 270, 297 и 327, включая так называемый Fc-мутант «DANA» с заменой остатков 265 и 297 аланином (патент США №7332581).
В другом аспекте Fc-домен представляет собой Fc-домен IgG4. Антитела IgG4 демонстрируют сниженную аффинность связывания с Fc-рецепторами и сниженные эффекторные функции по сравнению с антителами IgG1. В более конкретном аспекте Fc-домен представляет собой Fc-домен IgG4, содержащий аминокислотную замену в позиции S228 (нумерация Kabat), в частности, аминокислотную замену S228P. В более конкретном аспекте Fc-домен представляет собой Fc-домен IgG4, содержащий аминокислотные замены L235E, S228P и P329G (нумерация EU). Такие мутантные Fc-домены IgG4 и их свойства связывания Fcy-рецепторов также описаны в WO 2012/130831.
Мутантные Fc-домены можно получать с помощью аминокислотной делеции, замены, вставки или модификации, используя генетические и химические методы, хорошо известные в данной области техники. Генетические методы могут включать сайт-специфический мутагенез кодирующей последовательности ДНК, ПЦР, генный синтез и т.п. Правильные нуклеотидные изменения можно верифицировать, например, путем секвенирования.
Связывание с Fc-рецепторами можно легко определить, например, методом ELISA или методом поверхностного плазмонного резонанса (ППР), используя стандартное оборудование, такое как инструмент BIAcore (GE Healthcare), и Fc-рецепторы, которые могут быть получены методом рекомбинантной экспрессии. В альтернативном варианте аффинность связывания Fc-доменов или активирующих клетки антител, содержащих Fc-домен, с Fc-рецепторами можно оценить, используя линии клеток, которые экспрессируют конкретные Fc-рецепторы, такие как человеческие NK-клетки, экспрессирующие FcγIIIa-рецептор.
Эффекторную функцию Fc-домена или антигенсвязывающих молекул по изобретению, содержащих Fc-домен, можно измерить методами, известными в данной области техники. Подходящий анализ для измерения АЗКЦ описан в данном документе. Другие примеры in vitro анализов для оценки АЗКЦ-активности представляющей интерес молекулы описаны в патенте США №5500362; Hellstrom et al. Proc Natl Acad Sci USA 83, 7059-7063 (1986) и Hellstrom et al., Proc Natl Acad Sci USA 82, 1499-1502 (1985); патенте США №5821337; Bruggemann et al., J Exp Med 166, 1351-1361 (1987). В альтернативном варианте можно использовать нерадиоактивные методы анализа (смотрите, например, нерадиоактивный анализ цитотоксичности ACTI™ для проточной цитометрии (CellTechnology, Inc. Mountain View, CA); и нерадиоактивный анализ цитотоксичности CytoTox 96® (Promega, Madison, WI)). Эффекторные клетки, подходящие для такого анализа, включают мононуклеарные клетки периферической крови (МКПК) и естественные клетки-киллеры (NK). В альтернативном или дополнительном варианте АЗКЦ-активность представляющей интерес молекулы можно оценивать in vivo, например, в животной модели, такой как описана в Clynes et al., Proc Natl Acad Sci USA 95, 652-656 (1998).
В некоторых аспектах снижено связывание Fc-домена с компонентом комплемента, в частности с C1q. Соответственно, в некоторых аспектах, в которых Fc-домен сконструирован так, чтобы иметь сниженную эффекторную функцию, указанная сниженная эффекторная функция включает сниженную КЗЦ. Можно проводить анализ связывания C1q, чтобы определить, способны ли биспецифические антитела по изобретению связывать C1q и, следовательно, обладают ли они активностью КЗЦ. Смотрите, например, ИФА-анализ связывания C1q и С3с в WO 2006/029879 и WO 2005/100402. Для оценки активации комплемента можно проводить анализ КЗЦ (смотрите, например, Gazzano-Santoro et al., J Immunol Methods 202, 163 (1996); Cragg et al., Blood 101, 1045-1052 (2003); и Cragg and Glennie, Blood 103, 2738-2743 (2004)).
В одном конкретном аспекте Fc-домен, проявляющий сниженную аффинность связывания с Fc-рецептором и/или сниженную эффекторную функцию по сравнению с Fc-доменом нативного IgG1, представляет собой Fc-домен IgG1 человека, содержащий аминокислотные замены L234A, L235A и, необязательно, P329G, или Fc-домен IgG4 человека, содержащий аминокислотные замены S228P, L235E и, необязательно, P329G (нумерация в соответствии с индексом EU по Kabat). Конкретнее, он представляет собой Fc-домен IgG1 человека, содержащий аминокислотные замены L234A, L235A и P329G (нумерация в соответствии с индексом EU по Kabat).
Модификации Fc-домена, способствующие гетеродимеризации
Биспецифические агонистические CD28-антигенсвязывающие молекулы по изобретению содержат разные антигенсвязывающие сайты, слитые с одной или другой из двух субъединиц Fc-домена, таким образом, две субъединицы Fc-домена могут содержаться в двух неидентичных полипептидных цепях. Рекомбинантная коэкспрессия этих полипептидов и последующая димеризация приводит к нескольким возможным комбинациям двух полипептидов. Таким образом, чтобы повысить выход и чистоту биспецифических антигенсвязывающих молекул по изобретению при рекомбинантном получении, целесообразно внести в Fc-домен биспецифических антигенсвязывающих молекул по изобретению модификацию, способствующую ассоциации необходимых полипептидов.
Соответственно, в конкретных аспектах изобретение относится к биспецифической агонистической CD28-антигенсвязывающей молекуле с одновалентным связыванием с CD28, содержащей (а) один антигенсвязывающий домен, способный к специфическому связыванию с CD28, (б) по меньшей мере один антигенсвязывающий домен, способный к специфическому связыванию с опухолеассоциированным антигеном, и (в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию, при этом Fc-домен содержит модификацию, способствующую ассоциации первой и второй субъединиц Fc-домена. Сайтом наиболее активного белок-белкового взаимодействия между двумя субъединицами Fc-домена IgG человека является СН3-домен Fc-домена. Таким образом, в одном аспекте указанная модификация находится в СН3-домене Fc-домена.
В конкретном аспекте указанная модификация представляет собой так называемую модификацию типа «выступ во впадину», содержащую модификацию «выступа» в одной из двух субъединиц Fc-домена и модификацию «впадины» в другой из двух субъединиц Fc-домена. Таким образом, изобретение относится к биспецифической агонистической CD28-антигенсвязывающей молекуле с одновалентным связыванием с CD28, содержащей (а) один антигенсвязывающий домен, способный к специфическому связыванию с CD28, (б) по меньшей мере один антигенсвязывающий домен, способный к специфическому связыванию с опухолеассоциированным антигеном, и (в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию, при этом первая субъединица Fc-домена содержит выступы, а вторая субъединица Fc-домена содержит впадины в соответствии с методом «выступы во впадины». В конкретном аспекте первая субъединица Fc-домена содержит аминокислотные замены S354C и T366W (нумерация EU), а вторая субъединица Fc-домена содержит аминокислотные замены Y349C, T366S и Y407V (нумерация в соответствии с индексом EU по Kabat).
Технология «выступ-во-впадину» описана, например, в US 5731168; US 7695936; Ridgway et al., Prot Eng 9, 617-621 (1996) и Carter, J Immunol Meth 248, 7-15 (2001). В общем случае этот способ включает внесение выступа («узла») в контактную поверхность первого полипептида и соответствующей впадины («полости») в контактную поверхность второго полипептида, так что выступ может располагаться во впадине так, чтобы стимулировать образование гетеродимера и затруднять образование гомодимера. Выступы конструируют путем замены небольших аминокислотных боковых цепей в контактной поверхности первого полипептида на более крупные боковые цепи (например тирозин или триптофан). Компенсаторные «полости» идентичного или сходного размера с выступами создают в контактной поверхности второго полипептида путем замещения крупных аминокислотных боковых цепей меньшими (например, аланином или треонином).
Соответственно, в одном аспекте в СН3-домене первой субъединицы Fc-домена биспецифических антигенсвязывающих молекул по изобретению аминокислотный остаток замещен аминокислотным остатком, имеющим больший объем боковой цепи, тем самым создавая выступ в СН3-домене первой субъединицы, который совместим с полостью в СН3-домене второй субъединицы, а в СН3-домене второй субъединицы Fc-домена аминокислотный остаток замещен аминокислотным остатком, имеющим меньший объем боковой цепи, тем самым создавая впадину в СН3-домене второй субъединицы, в которой может размещаться выступ в СН3-домене первой субъединицы. Выступ и полость можно создавать путем изменения нуклеиновой кислоты, кодирующей полипептиды, например, с помощью сайт-специфического мутагенеза, или с помощью пептидного синтеза. В конкретном аспекте в СН3-домене первой субъединицы Fc-домена остаток треонина в позиции 366 замещен остатком триптофана (T366W), а в СН3-домене второй субъединицы Fc-домена остаток тирозина в позиции 407 замещен остатком валина (Y407V). В одном аспекте во второй субъединицы Fc-домена дополнительно остаток треонина в позиции 366 замещен остатком серина (T366S), а остаток лейцина в позиции 368 замещен остатком аланина (L368A).
В дополнительном аспекте в первой субъединице Fc-домена дополнительно остаток серина в позиции 354 замещен остатком цистеина (S354C), а во второй субъединице Fc-домена дополнительно остаток тирозина в позиции 349 замещен остатком цистеина (Y349C). Внесение этих двух остатков цистеина приводит к образованию дисульфидного мостика между двумя субъединицами Fc-домена, дополнительно стабилизируя димер (Carter (2001), J Immunol Methods 248, 7-15). В конкретном аспекте первая субъединица Fc-домена содержит аминокислотные замены S354C и T366W (нумерация EU), а вторая субъединица Fc-домена содержит аминокислотные замены Y349C, T366S и Y407V (нумерация в соответствии с индексом EU по Kabat).
В альтернативном аспекте модификация, способствующая ассоциации первой и второй субъединиц Fc-домена включает модификацию, которая опосредует эффекты электростатического взаимодействия, например, как описано в публикации РСТ WO 2009/089004. В общем случае этот способ включает замену одного или более аминокислотных остатков на поверхности контакта двух субъединиц Fc-домена заряженными аминокислотными остатками так, чтобы образование гомодимера становилось электростатически невыгодным, но гетеродимеризация была бы электростатический выгодной.
С-конец тяжелой цепи представленной в данном документе биспецифической агонистической CD28-антигенсвязывающей молекулы может представлять собой полный С-конец, оканчивающийся аминокислотными остатками PGK. С-конец тяжелой цепи может представлять собой укороченный С-конец, в котором были удалены один или два С-концевых аминокислотных остатка. В одном предпочтительном аспекте С-конец тяжелой цепи представляет собой укороченное С-концевое окончание Р. В одном предпочтительном аспекте С-конец тяжелой цепи представляет собой укороченное С-концевое окончание PG. В одном аспекте из всех представленных в данном документе аспектов CD28-антигенсвязывающая молекула, содержащая тяжелую цепь, содержащую С-концевой СН3-домен, описанный в данном документе, содержит С-концевой глицин-лизиновый дипептид (G446 и К447, нумерация в соответствии с индексом EU по Kabat). В одном аспекте из всех представленных в данном документе аспектов CD28-антигенсвязывающая молекула, содержащая тяжелую цепь, содержащую С-концевой СН3-домен, описанный в данном документе, содержит С-концевой остаток глицина (G446, нумерация в соответствии с индексом EU по Kabat).
Модификации в Fab-доменах
В одном аспекте изобретение относится к биспецифической агонистической CD28-антигенсвязывающей молекуле, характеризующейся одновалентным связыванием с CD28, содержащей (а) один антигенсвязывающий домен, способный к специфическому связыванию с CD28, (б) по меньшей мере один антигенсвязывающий домен, способный к специфическому связыванию с опухолеассоциированным антигеном, и (в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию, при этом по меньшей мере один антигенсвязывающий домен, способный к специфическому связыванию с опухолеассоциированным антигеном, представляет собой Fab-фрагмент, а в Fab-фрагменте вариабельные домены VH и VL или константные домены СН1 и CL обменены в соответствии с технологией Crossmab.
Мультиспецифические антитела с замещением/обменом доменов в одном связывающем плече (CrossMabVH-VL или CrossMabCH-CL) подробно описаны в WO2009/080252 и Schaefer, W. et al, PNAS, 108 (2011) 11187-1191. Они несомненно снижают количество побочных продуктов, возникающих в результате несовпадения легкой цепи против первого антигена с неправильной тяжелой цепью против второго антигена (по сравнению с подходами без такого обмена доменов).
В одном аспекте изобретение относится к биспецифической агонистической CD28-антигенсвязывающей молекуле, характеризующейся одновалентным связыванием с CD28, содержащей (а) один антигенсвязывающий домен, способный к специфическому связыванию с CD28, (б) по меньшей мере один антигенсвязывающий домен, способный к специфическому связыванию с опухолеассоциированным антигеном, и (в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию, при этом в Fab-фрагментах, способных к специфическому связыванию с опухолеассоциированным антигеном, константные домены CL и СН1 замещены друг другом так, что домен СН1 является частью легкой цепи, а домен CL является частью тяжелой цепи.
В другом аспекте и для дополнительного улучшения правильного спаривания биспецифическая агонистическая CD28-антигенсвязывающая молекула, характеризующаяся одновалентным связыванием с CD28, содержащая (а) один антигенсвязывающий домен, способный к специфическому связыванию с CD28, (б) по меньшей мере один антигенсвязывающий домен, способный к специфическому связыванию с опухолеассоциированным антигеном, и (в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию, может содержать разные заряженные аминокислотные замены (так называемые «заряженные остатки»). Эти модификации вносят в скрещенные или не скрещенный домены СН1 и CL. В конкретном аспекте изобретение относится к биспецифической агонистической CD28-антигенсвязывающей молекуле, в которой в одном из доменов CL аминокислота в позиции 123 (нумерация EU) была замещена аргинином (R) и аминокислота в позиции 124 (нумерация EU) была замещена лизином (K), и в которой в одном из доменов СН1 аминокислоты в позиции 147 (нумерация EU) и в позиции 213 (нумерация EU) были замещены глутаминовой кислотой (Е). В одном конкретном аспекте в домене CL Fab-фрагмента, способного к специфическому связыванию с CD28, аминокислота в позиции 123 (нумерация EU) была замещена аргинином (R) и аминокислота в позиции 124 (нумерация EU) была замещена лизином (K), и в домене СН1 Fab-фрагмента, способного к специфическому связыванию с CD28, аминокислоты в позиции 147 (нумерация EU) и в позиции 213 (нумерация EU) были замещены глутаминовой кислотой (Е).
Полинуклеотиды
В изобретении дополнительно предложены выделенные полинуклеотиды, кодирующие описанную в данному документе биспецифическую агонистическую CD28-антигенсвязывающую молекулу или ее фрагмент. Один или более выделенных полинуклеотидов, кодирующих биспецифическую агонистическую CD28-антигенсвязывающую молекулу по изобретению, можно экспрессировать в виде одного полинуклеотида, который кодирует всю антигенсвязывающую молекулу, или в виде нескольких (например, двух или более) коэкспрессируемых полинуклеотидов. Полипептиды, кодируемые коэкспрессируемыми полинуклеотидами, могут ассоциироваться, например, посредством дисульфидных связей или иным образом, с образованием функциональной антигенсвязывающей молекулы. Например, часть легкой цепи иммуноглобулина может кодироваться другим полинуклеотидом, чем часть тяжелой цепи иммуноглобулина. При коэкспрессии полипептиды тяжелой цепи будут ассоциироваться с полипептидами легкой цепи с образованием иммуноглобулина. В некоторых аспектах выделенный полинуклеотид кодирует всю биспецифическую агонистическую CD28-антигенсвязывающую молекулу в соответствии с изобретением, описанную в данном документе. В других аспектах выделенный полинуклеотид кодирует полипептид, содержащийся в биспецифической агонистической CD28-антигенсвязывающей молекуле в соответствии с изобретением, описанной в данном документе. В определенных аспектах полинуклеотид или нуклеиновая кислота представляет собой ДНК. В других аспектах полинуклеотид по настоящему изобретению представляет собой РНК, например, в форме матричной РНК (мРНК). РНК по настоящему изобретению может быть одноцепочечной или двухцепочечной. Рекомбинантные методы Биспецифические агонистические CD28-антигенсвязывающие молекулы по изобретению можно получать, например, посредством твердофазного пептидного синтеза (например, твердофазного синтеза Меррифильда) или рекомбинантного получения. В случае рекомбинантного получения один или более полинуклеотидов, кодирующих биспецифическую агонистическую CD28-антигенсвязывающую молекулу или ее полипептидные фрагменты, например, как описано выше, выделяют и вставляют в один или более векторов для последующего клонирования и/или экспрессии в клетке-хозяине. Такой полинуклеотид можно легко выделить и секвенировать, используя традиционные процедуры. В одном аспекте изобретения получают вектор, предпочтительно экспрессионный вектор, содержащий один или более полинуклеотидов по изобретению. Для конструирования экспрессионных векторов, содержащих кодирующую последовательность антитела (фрагмента) наряду с соответствующими сигналами управления транскрипцией/трансляцией, можно использовать методы, хорошо известные специалистам в данной области техники. Эти методы включают in vitro технологии рекомбинантных ДНК, технологии синтеза и in vivo рекомбинации/генетической рекомбинации. Смотрите, например, технологии, описанные в Maniatis et al., MOLECULAR CLONING: A LABORATORY MANUAL, Cold Spring Harbor Laboratory, N.Y. (1989); and Ausubel et al., CURRENT PROTOCOLS IN MOLECULAR BIOLOGY, Greene Publishing Associates and Wiley Interscience, N.Y. (1989). Экспрессионный вектор может быть частью плазмиды, вируса или может представлять собой фрагмент нуклеиновой кислоты. Экспрессионный вектор содержит экспрессионную кассету, в которую клонирован полинуклеотид, кодирующий антитело или его полипептидные фрагменты (т.е. кодирующая область), в функциональной ассоциации с промотором и/или другими элементами управления транскрипцией/трансляцией. В контексте данного документа «кодирующая область» представляет собой часть нуклеиновой кислоты, которая состоит из кодонов, транслируемых в аминокислоты. Хотя «стоп-кодон» (TAG, TGA или TAA) не транслируется в аминокислоту, он может считаться частью кодирующей области, в случае наличия, но любые фланкирующие последовательности, например, промоторы, сайты связывания рибосомы, терминаторы транскрипции, интроны, 5' и 3' нетранслируемые области и т.п., не являются частью кодирующей области. Две или более кодирующих областей могут присутствовать в одной полинуклеотидной конструкции, например, в одном векторе, или в разных полинуклеотидных конструкциях, например в отдельных (разных) векторах. Кроме того, любой вектор может содержать одну кодирующую область или может содержать две или более кодирующих областей, например, вектор по настоящему изобретению может кодировать один или более полипептидов, которые пост- или ко-трансляционно разделяются на конечные белки посредством протеолитического расщепления. Кроме того, вектор, полинуклеотид или нуклеиновая кислота по изобретению могут кодировать гетерологичные кодирующие области, слитые или не слитые с полинуклеотидом, кодирующим антитело по изобретению или его полипептидные фрагменты, или ее варианты или производные. Гетерологичные кодирующие области включают, без ограничения, специальные элементы или мотивы, такие как секреторный сигнальный пептид или гетерологичный функциональный домен. Функциональная ассоциация имеет место, когда кодирующая область для генного продукта, например, полипептида, связана с одной или более регуляторными последовательностями таким образом, чтобы экспрессия генного продукта была под управлением или контролем регуляторной(ых) последовательности(ей). Два фрагмента ДНК (такие как кодирующая полипептид область и связанный с ней промотор) «функционально связаны», если индукция функции промотора приводит к транскрипции мРНК, кодирующей необходимый генный продукт, и если природа связи между двумя фрагментами ДНК не препятствует способности экспрессионных регуляторных последовательностей управлять экспрессией генного продукта или не препятствует способности матрицы ДНК к транскрипции. Таким образом, промоторная область функционально связана с нуклеиновой кислотой, кодирующей полипептид, если промотор способен обеспечивать транскрипцию этой нуклеиновой кислоты. Промотор может представлять собой клеточно-специфический промотор, который обеспечивает существенную транскрипцию ДНК только в определенных клетках. Для управления клеточно-специфической транскрипцией с полинуклеотидом могут быть функционально связаны другие элементы управления транскрипцией помимо промотора, например, энхансеры, операторы, репрессоры и сигналы терминации транскрипции.
Подходящие промоторы и другие области управления транскрипцией описаны в данном документе. Различные области управления транскрипцией хорошо известны специалистам в данной области техники. Они включают, без ограничения, области управления транскрипцией, функциональные в клетках позвоночных, такие как, без ограничения, сегменты промоторов и энхансеров из цитомегаловирусов (например, немедленно ранний промотор в сочетании с интроном-А), вируса обезьян 40 (например, ранний промотор) и ретровирусов (таких как, например, вирус саркомы Рауса). Другие области управления транскрипцией включают полученные из генов позвоночных, таких как гены актина, белка теплового шока, бычьего гормона роста и кроличьего  -глобина, а также другие последовательности, способные управлять экспрессией генов в эукариотических клетках. Дополнительные области управления транскрипцией включают тканеспецифические промоторы и энхансеры, а также индуцибельные промоторы (например, промоторы, индуцируемые тетрациклинами). Аналогично, различные элементы управления трансляцией хорошо известны специалистам в данной области техники. Они включают, но не ограничиваются этим, сайты связывания рибосом, кодоны инициации и терминации трансляции и элементы, полученные из вирусных систем (в частности, сайт внутренней посадки рибосомы или IRES, также называемый последовательностью CITE). Экспрессионная кассета также может содержать другие элементы, такие как точка начала репликации и/или элементы интеграции хромосомы, такие как ретровинусные длинные концевые повторы (ДКП) или инвертированные концевые повторы (ИКП) аденоассоциированного вируса (AAV).
-глобина, а также другие последовательности, способные управлять экспрессией генов в эукариотических клетках. Дополнительные области управления транскрипцией включают тканеспецифические промоторы и энхансеры, а также индуцибельные промоторы (например, промоторы, индуцируемые тетрациклинами). Аналогично, различные элементы управления трансляцией хорошо известны специалистам в данной области техники. Они включают, но не ограничиваются этим, сайты связывания рибосом, кодоны инициации и терминации трансляции и элементы, полученные из вирусных систем (в частности, сайт внутренней посадки рибосомы или IRES, также называемый последовательностью CITE). Экспрессионная кассета также может содержать другие элементы, такие как точка начала репликации и/или элементы интеграции хромосомы, такие как ретровинусные длинные концевые повторы (ДКП) или инвертированные концевые повторы (ИКП) аденоассоциированного вируса (AAV).
Кодирующие области полинуклеотидов и нуклеиновых кислот по настоящему изобретению могут быть связаны с дополнительными кодирующими областями, которые кодируют секреторные или сигнальные пептиды, которые управляют секрецией полипептида, кодируемого полинуклеотидом по настоящему изобретению. Например, если необходима секреция антитела или его полипептидных фрагментов, ДНК, кодирующую сигнальную последовательность, можно помещать выше нуклеиновой кислоты, кодирующей антитело по изобретению или его полипептидные фрагменты. В соответствии с сигнальной гипотезой, белки, секретируемые клетками млекопитающих, имеют сигнальный пептид или секреторную лидерную последовательность, которые отщепляются от зрелого белка после инициации экспорта растущей белковой цепи через шероховатый эндоплазматический ретикулум. Специалистам в данной области техники известно, что полипептиды, секретируемые клетками позвоночных, в общем случае имеют сигнальный пептид, слитый с N-концом полипептида, который отщепляется от транслируемого полипептида с получением секретируемой или «зрелой» формы полипептида. В определенных вариантах осуществления используют нативный сигнальный пептид, например, сигнальный пептид тяжелой цепи или легкой цепи иммуноглобулина, или функциональное производное этой последовательности, которое сохраняет способность управлять секрецией полипептида, который с ней функционально связан. В альтернативном варианте можно использовать гетерологичный сигнальный пептид млекопитающего или его функциональное производное. Например, лидерную последовательность дикого типа можно заменить лидерной последовательностью человеческого тканевого активатора плазминогена (ТРА) или мышиной β-глюкуронидазы.
ДНК, кодирующая короткую белковую последовательность, которую можно использовать для облегчения очистки (например, гистидиновый тэг) или мечения биспецифической агонистической CD28-антигенсвязывающей молекулы, может быть включена в середине или в концах полинуклеотида, кодирующего антитело по изобретению или его полипептидные фрагменты.
В дополнительном аспекте изобретения предложена клетка-хозяин, содержащая один или более полинуклеотидов по изобретению. В определенных вариантах осуществления предложена клетка-хозяин, содержащая один или более векторов по изобретению. Полинуклеотиды и векторы могут содержать любые фрагменты, отдельно или в комбинации, описанные в данном документе в отношение полинуклеотидов и векторов, соответственно. В одном аспекте клетка-хозяин содержит вектор (например, была трансформирована или трансфицирована им), содержащий полинуклеотид, который кодирует антитело по изобретению (или его часть). В контексте данного документа термин «клетка-хозяин» относится к любому типу клеточной системы, которую можно сконструировать для создания слитых белков по изобретению или их фрагментов. Клетки-хозяева, подходящие для репликации и поддержания экспрессии антигенсвязывающих молекул, хорошо известны в данной области техники. Такие клетки можно трансфицировать или трансдуцировать, сообразно ситуации, конкретным экспрессионным вектором, и можно выращивать большие количества содержащих вектор клеток для засевания крупномасштабных ферментеров для получения достаточных количеств антигенсвязывающей молекулы для клинических применений. Подходящие клетки включают прокариотические микроорганизмы, такие как Е. coli, или различные эукариотические клетки, такие как клетки яичника китайского хомяка (СНО), клетки насекомых и т.п. Например, полипептиды можно получать в бактериях, в частности, если не требуется гликозилирование. После экспрессии полипептид можно выделять из бактериальной клеточной пасты в растворимую фракцию и дополнительно очищать. Помимо прокариот, эукариотические микробы, такие как нитевидные грибы или дрожжи, являются подходящими хозяевами для клонирования или экспрессии кодирующих полипептиды векторов, включая штаммы грибов и дрожжей, пути гликозилирования которых были «гуманизированы», что приводит к получению полипептида с частично или полностью человеческим профилем гликозилирования. Смотрите Gerngross, Nat Biotech 22, 1409-1414 (2004), и Li et al., Nat Biotech 24, 210-215 (2006).
Клетки-хозяев, подходящие для экспрессии (гликозилированных) полипептидов, также получают из многоклеточных организмов (беспозвоночных и позвоночных). Примеры клеток беспозвоночных включают клетки растений и насекомых. Были идентифицированы многочисленные бакуловирусные штаммы, которые можно использовать в сочетании с клетками насекомых, в частности, для трансфекции клеток Spodoptera frugiperda. Культуры клеток растений также можно использовать в качестве хозяев. Смотрите, например, патенты США №№5959177, 6040498, 6420548, 7125978 и 6417429 (в которых описана технология PLANTIBODIES™ для получения антител в трансгенных растениях). Клетки позвоночных также можно использовать в качестве хозяев. Например, можно использовать линии клеток млекопитающих, которые адаптированы для роста в суспензии. Другими примерами применимых линий клеток-хозяев млекопитающих являются линия клеток почки обезьяны CV1, трансформированная SV40 (COS-7); линия клеток почки эмбриона человека (клетки 293 или 293Т, описанные, например, в in Graham et al., J Gen Virol 36, 59 (1977)), клетки почки новорожденного хомяка (ВНК); клетки Сертоли мышей (клетки ТМ4, описанные, например, в Mather, Biol Reprod 23, 243-251 (1980)), клетки почки обезьяны (CV1), клетки почки африканской зеленой мартышки (VERO-76), клетки карциномы шейки матки человека (HELA), клетки почки собаки (MDCK), клетки печени серой крысы (BRL ЗА), клетки легкого человека (W138), клетки печени человека (Hep G2), клетки опухоли молочной железы мышей (ММТ 060562), клетки TRI (описанные, например, в Mather et al., Annals N.Y. Acad Sci 383, 44-68 (1982)), клетки MRC 5 и клетки FS4. Другие применимые линии клеток-хозяев млекопитающих включают клетки яичника китайского хомяка (СНО), включая dhfr- клетки СНО (Urlaub et al., Proc Natl Acad Sci USA 77, 4216 (1980)); и линии клеток миеломы, такие как YO, NS0, РЗХ63 и Sp2/0. Обзор некоторых линий клеток-хозяев млекопитающих, подходящих для выработки белка, смотрите, например, в Yazaki and Wu, Methods in Molecular Biology, Vol. 248 (B.K.C. Lo, ed., Humana Press, Totowa, NJ), pp.255-268 (2003). Клетки-хозяева включают культивируемые клетки, например, культивируемые клетки млекопитающих, клетки дрожжей, клетки насекомых, клетки бактерий и клетки растений, если называть лишь некоторые, но также клетки, принадлежащие трансгенному животному, трансгенному растению или культивируемой ткани растения или животного. В одном варианте осуществления клетка-хозяин представляет собой эукариотическую клетку, предпочтительно клетку млекопитающего, такую как клетка яичника китайского хомяка (СНО), клетка почки эмбриона человека (HEK) или лимфоидная клетка (например, клетка Y0, NS0, Sp20). Стандартные технологии для экспрессии чужеродных генов в этих системах известны в данной области техники. Клетки, экспрессирующие полипептид, содержащий тяжелую или легкую цепь иммуноглобулина, можно конструировать так, чтобы они также экспрессировали другую цепь иммуноглобулина, чтобы экспрессируемый продукт представлял собой иммуноглобулин, который имеет как тяжелую, так и легкую цепь.
В одном аспекте предложен способ получения биспецифической агонистической CD28-антигенсвязывающей молекулы по изобретению или ее полипептидных фрагментов, включающий культивирование клетки-хозяина, содержащей полинуклеотиды, кодирующие антитело по изобретению или его полипептидные фрагменты, предложенные в данном документе, в условиях, подходящих для экспрессии антитела по изобретению или его полипептидных фрагментов, и выделение антитела по изобретению или его полипептидных фрагментов из клетки-хозяина (или культуральной среды клеток-хозяев).
В определенных аспектах фрагменты, способные к специфическому связыванию с антигеном клетки-мишени (например, Fab-фрагменты), образующие часть антигенсвязывающей молекулы, содержат по меньшей мере одну вариабельную область иммуноглобулина, способную связываться с антигеном. Вариабельные области могут образовывать часть и быть полученными из встречающихся в природе или не встречающихся в природе антител и их фрагментов. Способы получения поликлональных антител и моноклональных антител хорошо известны в данной области техники (смотрите, например, Harlow and Lane, "Antibodies, a laboratory manual", Cold Spring Harbor Laboratory, 1988). He встречающиеся в природе антитела можно конструировать, используя твердофазный пептидный синтез, можно получать рекомбинантно (например, как описано в патенте США №4186567) или можно получать, например, посредством скрининга комбинаторных библиотек, содержащих вариабельные тяжелые цепи и вариабельные легкие цепи (смотрите, например, патент США №5969108 авторства McCafferty).
В изобретении можно использовать иммуноглобулин любых видов животных. Неограничивающие примеры иммуноглобулинов, применимых в настоящем изобретении, могут быть получены от мышей, приматов или человека. Если слитый белок предназначен для применения человеком, можно использовать химерную форму иммуноглобулина, в которой константные области иммуноглобулина получены от человека. Гуманизированные или полностью человеческие формы иммуноглобулина также можно получать в соответствии со способами, хорошо известными в данной области техники (смотрите, например, патент США №5565332 авторства Winter). Гуманизацию можно обеспечивать различными способами, включая, но не ограничиваясь этим, (а) прививание нечеловеческих (например, донорного антитела) CDR в человеческие (например, реципиентного антитела) каркасную область и константные области с сохранением или нет критически важных каркасных остатков (например, тех, которые важны для сохранения хороших антигенсвязывающей аффинности или функций антитела), (б) прививание только нечеловеческих определяющих специфичность областей (SDR или a-CDR; остатки, критически важные для взаимодействия антиген антитело) в человеческие каркасную область и константные области, или (в) трансплантацию всех нечеловеческих вариабельных доменов, но с «маскировкой» их человекоподобным участком путем замещения поверхностных остатков. Гуманизированные антитела и способы их получения описаны, например, в Almagro and Fransson, Front Biosci 13, 1619-1633 (2008), и дополнительно описаны, например, в Riechmann et al., Nature 332, 323-329 (1988); Queen et al., Proc Natl Acad Sci USA 86, 10029-10033 (1989); патентах США №№5821337, 7527791, 6982321 и 7087409; Jones et al., Nature 321, 522-525 (1986); Morrison et al., Proc Natl Acad Sci 81, 6851-6855 (1984); Morrison and Oi, Adv Immunol 44, 65-92 (1988); Verhoeyen et al., Science 239, 1534-1536 (1988); Padlan, Molec Immun 31(3), 169-217 (1994); Kashmiri et al., Methods 36, 25-34 (2005) (где описано прививание SDR (a-CDR)); Padlan, Mol Immunol 28, 489-498 (1991) (где описано «изменение поверхности»); Dall'Acqua et al., Methods 36, 43-60 (2005) (где описана «перетасовка FR»); и Osbourn et al., Methods 36, 61-68 (2005) и Klimka et al., Br J Cancer 83, 252-260 (2000) (где описан подход «направленного отбора» для перетасовки FR). Конкретными иммуноглобулинами в соответствии с изобретением являются иммуноглобулины человека. Человеческие антитела и человеческие вариабельные области можно получать, используя различные технологии, известные в данной области техники. Человеческие антитела в целом описаны в van Dijk and van de Winkel, Curr Opin Pharmacol 5, 368-74 (2001) и Lonberg, Curr Opin Immunol 20, 450-459 (2008). Человеческие вариабельные области могут образовывать часть и могут быть получены из человеческих моноклональных антител, созданных методом гибридомы (смотрите, например, Monoclonal Antibody Production Techniques and Applications, pp. 51-63 (Marcel Dekker, Inc., New York, 1987)). Человеческие антитела и человеческие вариабельные области можно получать путем введения иммуногена трансгенному животному, модифицированному для выработки интактных человеческих антител или интактных антител с человеческими вариабельными областями в ответ на антигенную стимуляцию (смотрите, например, Lonberg, Nat Biotech 23, 1117-1125 (2005). Человеческие антитела и человеческие вариабельные области также можно создавать путем выделения последовательностей вариабельной области Fv-клона, отобранных из человеческих библиотек фагового дисплей (смотрите, например, Hoogenboom et al. в Methods in Molecular Biology 178, 1-37 (O'Brien et al., ed., Human Press, Totowa, NJ, 2001); и McCafferty et al., Nature 348, 552-554; Clackson et al., Nature 352, 624-628 (1991)). Как правило, фаг представляет фрагменты антител в виде одноцепочечных Fv-фрагментов (scFv) или Fab-фрагментов.
В определенных аспектах биспецифические агонистические CD28-антигенсвязывающие молекулы сконструированы так, чтобы они имели повышенную аффинность связывания, в соответствии, например, с методами, описанными в публикации РСТ WO 2012/020006 (смотрите примеры, относящиеся к созреванию аффинности) или публикацию заявки на патент США №2004/0132066. Способность антигенсвязывающих молекул по изобретению связываться с конкретной антигенной детерминантой можно определить с помощью ферментного иммуносорбентного анализа (ELISA) или других методик, известных специалисту в данной области техники, например, метода поверхностного плазмонного резонанса (Liljeblad et al., Glyco J 17, 323-329 (2000)) и традиционных анализов связывания (Heeley, Endocr Res 28, 217-229 (2002)). Для идентификации антигенсвязывающей молекулы, которая конкурирует с референсным антителом за связывание с конкретным антигеном, можно использовать конкурентный анализ. В определенных вариантах осуществления такая конкурирующая антигенсвязывающая молекула связывается с тем же эпитопом (например, линейным или конформационным эпитопом), который связывается референсной антигенсвязывающей молекулой. Подробное описание типовых методов картирования эпитопа, с которым связывается антигенсвязывающая молекула, приведены в Morris (1996) "Epitope Mapping Protocols", в Methods in Molecular Biology vol. 66 (Humana Press, Totowa, NJ). В иллюстративном конкурентном анализе иммобилизованный антиген инкубируют в растворе, содержащем первую меченную антигенсвязывающую молекулу, которая связывается с антигеном, и вторую немеченную антигенсвязывающую молекулу, которую исследуют в отношении ее способности конкурировать с первой антигенсвязывающей молекулой за связывание с антигеном. Вторая антигенсвязывающая молекула может находиться в гибридомном супернатанте. В качестве контроля иммобилизованный антиген инкубируют в растворе, содержащем первую меченную антигенсвязывающую молекулу, но не вторую немеченную антигенсвязывающую молекулу. После инкубации в условиях, обеспечивающих связывание первого антитела с антигеном, удаляют избыток несвязанного антитела и измеряют количество метки, связанной с иммобилизованным антигеном. Если количество метки, связанной с иммобилизованным антигеном, существенно снижено в исследуемом образце по сравнению с контрольным образцом, это указывает на то, что вторая антигенсвязывающая молекула конкурирует с первой антигенсвязывающей молекулой за связывание с антигеном. Смотрите Harlow and Lane (1988) Antibodies: A Laboratory Manual ch. 14 (Cold Spring Harbor Laboratory, Cold Spring Harbor, NY).
Биспецифические агонистические CD28-антигенсвязывающие молекулы по изобретению, полученные, как описано в данном документе, можно очищать известными в данной области техники методами, такими как высокоэффективная жидкостная хроматография, ионообменная хроматография, гель-электрофорез, аффинная хроматография, эксклюзионная хроматография и т.п. Фактические условия, применяемые для очистки конкретного белка, будут зависеть частично от факторов, таких как суммарный заряд, гидрофобность, гидрофильность и т.д., и будут очевидны для специалистов в данной области техники. Для очистки методом аффинной хроматографии можно использовать антитело, лиганд, рецептор или антиген, с которыми связывается антигенсвязывающая молекула. Например, очистки антигенсвязывающих молекул по изобретению методом аффинной хроматографии можно использовать матрицу с протеином А или протеином G. Последовательные этапы аффинной хроматографии с протеином А или G и эксклюзионной хроматографии можно использовать для выделения антигенсвязывающей молекулы по существу так, как описано в примерах. Чистоту CD28-антигенсвязывающей молекулы по изобретению или ее фрагментов можно определять любым из ряда хорошо известных аналитических методов, включая электрофорез, жидкостную хроматографию высокого давления и т.п. Например, было показано, что CD28-антигенсвязывающая молекула, экспрессируемая, как описано в примерах, является интактной и имеет надлежащую сборку, что было продемонстрировано восстанавливающим и невосстанавливающим ДСН-ПААГ.
Методы анализа
Идентификацию, скрининг или изучение характеристик предложенных в данном документе биспецифических агонистических CD28-антигенсвязывающих молекул в отношении их физических/химических свойств и/или биологической активности можно проводить с помощью различных методов анализа, известных в данной области техники.
1. Анализ аффинности
Аффинность предложенной в данном документе антигенсвязывающей молекулы в отношении соответствующей мишени можно определять в соответствии со способами, приведенными в примерах, методом поверхностного плазмонного резонанса (ППР), используя стандартный инструментарий, такой как инструмент Proteon (Bio-rad), и рецепторы или белки-мишени, которые можно получить путем рекомбинантной экспрессии. Аффинность содержащей тримерный лиганд семейства TNF антигенсвязывающей молекулы в отношении антигена клетки-мишени также можно определять методом поверхностного плазмонного резонанса (ППР), используя стандартный инструментарий, такой как инструмент Proteon (Bio-rad), и рецепторы или белки-мишени, которые можно получить путем рекомбинантной экспрессии. Конкретный иллюстративный и типовой вариант осуществления для определения аффинности связывания описан в примере 4. В соответствии с одним аспектом Kd измеряют методом поверхностного плазмонного резонанса, используя прибор Proteon® (Bio-Rad) при 25°С.
2. Методы анализа связывания и другие методы анализа
Связывание предложенной в данном документе биспецифической антигенсвязывающей молекулы с экспрессирующими соответствующий рецептор клетками можно оценивать, используя линии клеток, экспрессирующие конкретный рецептор или антиген-мишень, например, методом проточной цитометрии (FACS). В одном аспекте в анализе связывания используют клетки СНО, экспрессирующие CD28 человека (родительская линия клеток CHO-k1 АТСС №CCL-61, модифицированная для стабильной экспрессии CD28 человека).
В дополнительном аспекте использовали линии раковых клеток, экспрессирующие антиген клетки-мишени, например, FAP или CEA, CD19 или CD79b, чтобы продемонстрировать связывание биспецифических антигенсвязывающих молекул с антигеном клетки-мишени.
3. Методы анализа активности
В одном аспекте предложены методы анализа для идентификации CD28-антигенсвязывающих молекул, обладающих биологической активностью. Биологическая активность может включать, например, пролиферацию Т-клеток и секрецию цитокинов, измеряемые способом, описанным в примере 6, или уничтожение опухолевых клеток, измеряемое в примере 7. Также предложены антитела, обладающие такой биологической активностью in vivo и/или in vitro.
Фармацевтические композиции, составы и пути введения
В дополнительном аспекте в изобретении предложены фармацевтические композиции, содержащие любую из предложенных в данном документе биспецифических агонистических CD28-антигенсвязывающих молекул, например, для применения в любом из нижеприведенных терапевтических способов. В одном варианте осуществления фармацевтическая композиция содержит предложенную в данном документе биспецифическую агонистическую CD28-антигенсвязывающую молекулу и по меньшей мере один фармацевтически приемлемый эксципиент. В другом аспекте фармацевтическая композиция содержит предложенную в данном документе биспецифическую агонистическую CD28-антигенсвязывающую молекулу и по меньшей мере один дополнительный терапевтический агент, например, как описано ниже.
Фармацевтические композиции по настоящему изобретению содержат терапевтически эффективное количество одной или более биспецифических антигенсвязывающих молекул, растворенных или диспергированных в фармацевтически приемлемом эксципиенте. Выражения «фармацевтически или фармакологически приемлемый» относятся к молекулярным веществам и композициям, которые в общем случае являются нетоксичными для реципиентов в применяемых дозировках и концентрациях, т.е. не вызывают нежелательную, аллергическую или другую неблагоприятную реакцию при введении животному, такому как, например, человек, в зависимости от ситуации. Изготовление фармацевтической композиции, которая содержит по меньшей мере одну биспецифическую агонистическую CD28-антигенсвязывающую молекулу и, необязательно, дополнительный активный ингредиент, станет понятно специалистам в данной области техники в свете настоящего изобретения, и как проиллюстрировано в Remington's Pharmaceutical Sciences, 18th Ed. Mack Printing Company, 1990, включенной в данный документ посредством ссылки. В частности, композиции представляют собой лиофилизированные составы или водные растворы. В контексте данного документа «фармацевтически приемлемое вспомогательное вещество» включает любые и все растворители, буферы, дисперсионные среды, покрытия, поверхностно-активные вещества, антиоксиданты, консерванты (например, антибактериальные агенты, противогрибковые агенты), изотонические агенты, соли, стабилизаторы и их комбинации, как известно специалисту в данной области техники.
Парентеральные композиции включают разработанные для введения путем инъекции, например, подкожной, интрадермальной, внутриочаговой, внутривенной, внутриартериальной, внутримышечной, интратекальной или внутрибрюшинной инъекции. Для инъекции содержащие тримерный лиганд семейства TNF антигенсвязывающие молекулы по изобретению могут быть составлены в виде водных растворов, предпочтительно в физиологически совместимых буферах, таких как раствор Хэнкса, раствор Рингера или физиологический солевой буфер. Раствор может содержать вспомогательные агенты, такие как суспендирующие, стабилизирующие и/или диспергирующие агенты. В альтернативном варианте биспецифическая агонистическая CD28-антигенсвязывающая молекула может находиться в форме порошка для смешивания с подходящим носителем, например, стерильной апирогенной водой, перед применением. Стерильные инъекционные растворы готовят путем смешивания слитых белков по изобретению в необходимом количестве в подходящем растворителе с различными перечисленными выше другими ингредиентами в случае необходимости. Стерильность можно легко обеспечить, например, путем фильтрации через стерильные фильтровальные мембраны. В общем случает дисперсии готовят путем включения различных стерилизованных активных ингредиентов в стерильный носитель, который содержит основную дисперсионную среду и/или другие ингредиенты. В случае стерильных порошков для изготовления стерильных инъекционных растворов, суспензий или эмульсий предпочтительными способами изготовления являются технологии вакуумной сушки или сублимационной сушки, которые позволяют получать порошок активного ингредиента плюс любой дополнительный необходимый активный ингредиент из стерильно профильтрованной перед этим жидкой среды. Жидкую среду, при необходимости, нужно должным образом забуферить, а жидкий разбавитель сначала сделать изотоничным перед инъекцией с помощью достаточного количества солевого раствора или глюкозы. Композиция должна быть стабильной в условиях производства и хранения, и защищенной от загрязняющего действия микроорганизмов, таких как бактерии и грибы. Следует понимать, что загрязнение эндотоксинами необходимо свести к минимуму и поддерживать на безопасном уровне, например, менее 0,5 нг/мг белка. Подходящие фармацевтически приемлемые вспомогательные вещества включают, но не ограничиваются этим: буферы, такие как фосфат, цитрат и другие органические кислоты; антиоксиданты, включая аскорбиновую кислоту и метионин; консерванты (такие как хлорид октадецилдиметилбензил аммония; хлорид гексаметония; хлорид бензалкония; хлорид бензетония; фенол; бутиловый или бензиловый спирт; алкилпарабены, такие как метил- или пропилпарабен; катехол; резорцин; циклогексанол; 3-пентанол; и м-крезол); низкомолекулярные (содержащие менее 10 остатков) полипептиды; белки, такие как сывороточный альбумин, желатин или иммуноглобулины; гидрофильные полимеры, такие как поливинилпирролидон; аминокислоты, такие как глицин, глутамин, аспарагин, гистидин, аргинин или лизин; моносахариды, дисахариды и другие углеводы, включая глюкозу, маннозу или декстрины; хелатирующие агенты, такие как ЭДТА; сахара, такие как сахароза, маннит, трегалоза или сорбит; солеобразующие противоионы, такие как натрий; металлсодержащие комплексы (например, комплексы Zn-белок) и/или неионные поверхностно-активные вещества, такие как полиэтиленгликоль (ПЭГ). Водные инъекционные суспензии могут содержать соединения, которые увеличивают вязкость суспензии, такие как карбоксиметилцеллюлоза натрия, сорбит, декстран и т.п. Необязательно, суспензия может также содержать подходящие стабилизаторы или вещества, которые повышают растворимость соединений, для обеспечения получения высококонцентрированных растворов. Кроме того, суспензии активных соединений могут быть изготовлены в виде подходящих масляных инъекционных суспензий. Подходящие липофильные растворители или носители включают жирные масла, такие как кунжутное масло, или синтетические сложные эфиры жирных кислот, такие как этилолеаты или триглицериды, или липосомы.
Активные ингредиенты также могут быть заключены в микрокапсулах, полученных, например, с помощью методик коацервации или путем межфазной полимеризации, например, гидроксиметилцеллюлозные или желатиновые микрокапсулы и полиметилметакрилатные микрокапсулы, соответственно, в коллоидных системах для доставки лекарственных средств (например, липосомах, альбуминовых микросферах, микроэмульсиях, наночастицах и нанокапсулах) или в макроэмульсиях. Такие методики описаны в Remington's Pharmaceutical Sciences (18th Ed. Mack Printing Company, 1990). Можно получать препараты с замедленным высвобождением. Подходящие примеры препаратов с замедленным высвобождением включают полупроницаемые матрицы из твердых гидрофобных полимеров, содержащие полипептид, причем матрицы находятся в виде формованных изделий, например, пленок или микрокапсул. В конкретных вариантах осуществления длительное всасывание инъекционных композиций может быть достигнуто путем использования в композициях агентов, замедляющих всасывание, таких как, например, моностеарат алюминия, желатин или их комбинации. Типовые фармацевтически приемлемые вспомогательные вещества по данному документу дополнительно включают агенты для интерстициального диспергирования лекарственных средств, такие как растворимые нейтрально-активные гликопротеины гиалуронидазы (sHASEGP), например, человеческие растворимые гликопротеины гиалуронидазы РН-20, такие как rHuPH20 (HYLENEX®, Baxter International, Inc.). Некоторые типовые sHASEGP и способы их применения, включая rHuPH20, описаны в патентных публикациях США №№2005/0260186 и 2006/0104968. В одном аспекте sHASEGP комбинируют с одной или более дополнительными гликозаминогликаназами, такими как хондроитиназы. Типовые лиофилизированные составы антител описаны в патенте США №6267958. Водные составы антител включают описанные в патенте США №6171586 и WO2006/044908, причем последние составы содержат гистидин-ацетатный буфер. Помимо ранее описанных композиций биспецифическую агонистическую CD28-антигенсвязывающую молекулу также можно составлять в виде депо-препаратов. Такие составы пролонгированного действия можно вводить путем имплантации (например, подкожно или внутримышечно) или путем внутримышечной инъекции. Таким образом, например, биспецифическую агонистическую CD28-антигенсвязывающую молекулу можно смешивать с подходящими полимерными или гидрофобными материалами (например, в виде эмульсии в приемлемом масле) или ионообменными смолами, или в виде умеренно растворимых производных, например, умеренно растворимой соли.
Фармацевтические композиции, содержащие биспецифическую агонистическую CD28-антигенсвязывающую молекулу по изобретению, можно готовить посредством традиционных процессов смешивания, растворения, эмульсификации, инкапсуляции, захвата или лиофилизации. Фармацевтические композиции можно готовить традиционным образом, используя один или более физиологически приемлемых носителей, разбавителей или вспомогательных агентов, которые облегчают процесс обработки белков в препараты, которые можно использовать фармацевтически. Надлежащий состав зависит от выбранного пути введения. Биспецифическую агонистическую CD28-антигенсвязывающую молекулу по изобретению можно вмешивать в композицию в форме свободной кислоты или свободного основания, в нейтральной или солевой форме. Фармацевтически приемлемые соли представляют собой соли, которые в значительной степени сохраняют биологическую активность свободной кислоты или свободного основания. Они включают соли присоединения кислот, например, образуемые со свободными аминогруппами белковой композиции или образуемые с неорганическими кислотами, такими как, например, хлористоводородная или фосфорная кислоты, или такими органическими кислотами, как уксусная, щавелевая, винная или миндальная кислота. Соли, образуемые со свободными карбоксильными группами, также можно получать из неорганических оснований, таких как, например, гидроксиды натрия, калия, аммония, кальция или железа; или таких органических оснований, как изопропиламин, триметиламин, гистидин или прокаин. Фармацевтические соли, как правило, более растворимы в водных и других протонных растворителях, чем соответствующие формы свободных оснований. Композиция по данному документу также может содержать более одного активного ингредиента, если это необходимо для конкретного показания, подлежащего лечению, предпочтительно те, которые обладают дополняющими видами активности и не оказывают отрицательного влияния друг на друга. Такие активные ингредиенты приемлемо присутствуют в комбинации в количествах, которые эффективны для предполагаемой цели. Составы, предназначенные для введения in vivo, в общем случае являются стерильными. Стерильность можно легко обеспечить, например, путем фильтрации через стерильные фильтровальные мембраны.
Терапевтические способы и композиции
Любые биспецифические агонистические CD28-антигенсвязывающие молекулы, предложенные в данном документе, можно применять в терапевтических способах, отдельно или в комбинации.
В одном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула для применения в качестве лекарственного средства. В дополнительном аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула для применения в лечении рака. В определенных аспектах предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула для применения в способе лечения. В определенных аспектах в данном документе предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула для применения в способе лечения субъекта, имеющего рак, включающем введение субъекту эффективного количества биспецифической агонистической CD28-антигенсвязывающей молекулы. В одном таком варианте реализации способ дополнительно включает введение субъекту эффективного количества по меньшей мере одного дополнительного терапевтического агента.
В одном аспекте биспецифическая агонистическая CD28-антигенсвязывающая молекула предназначена для применения в лечении В-клеточного пролиферативного нарушения. В конкретных аспектах биспецифическая агонистическая CD28-антигенсвязывающая молекула предназначена для применения в лечении В-клеточного пролиферативного нарушения, выбранного из группы, состоящей из неходжкинской лимфомы (НХЛ), острого лимфоцитарного лейкоза (ОЛЛ), хронического лимфоцитарного лейкоза (ХЛЛ), диффузной В-крупноклеточной лимфомы (ДВКЛ), филликулярной лимфомц (ФЛ), мантийноклеточной лимфому (МКЛ), лимфомы маргинальной зоны (ЛМЗ), множественной миеломы (ММ) и лимфомы Ходжкина (ЛХ). В одном конкретном аспекте В-клеточный рак представляет собой неходжкинскую лимфому или острый лимфобластный лейкоз. В определенных аспектах предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула для применения в способе лечения. В определенных аспектах в данном документе предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула для применения в способе лечения субъекта, имеющего рак, включающем введение субъекту эффективного количества биспецифической агонистической CD28-антигенсвязывающей молекулы. В другом аспекте предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула для применения в способе лечения субъекта, имеющего В-клеточное пролиферативное нарушение, в частности В-клеточное пролиферативное нарушение, выбранное из группы, состоящей из неходжкинской лимфомы (НХЛ), острого лимфоцитарного лейкоза (ОЛЛ), хронического лимфоцитарного лейкоза (ХЛЛ), диффузной В-крупноклеточной лимфомы (ДВКЛ), филликулярной лимфомц (ФЛ), мантийноклеточной лимфому (МКЛ), лимфомы маргинальной зоны (ЛМЗ), множественной миеломы (ММ) и лимфомы Ходжкина (ЛХ), включающем введение субъекту эффективного количества биспецифической агонистической CD28-антигенсвязывающей молекулы. В одном таком варианте реализации способ дополнительно включает введение субъекту эффективного количества по меньшей мере одного дополнительного терапевтического агента.
В дополнительном аспекте предложена описанная в данном документе биспецифическая агонистическая CD28-антигенсвязывающая молекула для применения в иммунотерапии рака. В определенных вариантах осуществления предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула для применения в способе иммунотерапии рака. «Субъект» в соответствии с любым из вышеприведенных аспектов предпочтительно является человеком.
В дополнительном аспекте в данном документе предложено применение описанной в данном документе биспецифической агонистической CD28-антигенсвязывающей молекулы в производстве или изготовлении лекарственного средства. В одном варианте осуществления лекарственное средство предназначено для лечения рака. В дополнительном аспекте лекарственное средство предназначено для применения в способе лечения рака, включающем введение субъекту, имеющему рак, эффективного количества лекарственного средства. В одном таком аспекте указанный способ дополнительно включает введение субъекту эффективного количества по меньшей мере одного дополнительного терапевтического агента, например, как описано ниже. В другом аспекте лекарственное средство предназначено для лечения В-клеточного пролиферативного нарушения. В дополнительном аспекте лекарственное средство предназначено для применения в способе лечения рака или В-клеточного пролиферативного нарушения, включающем введение субъекту, имеющему рак, эффективного количества лекарственного средства.
В дополнительном аспекте в данном документе предложен способ лечения рака. В одном аспекте способ включает введение субъекту, имеющему рак, эффективного количества биспецифической агонистической CD28-антигенсвязывающей молекулы. В одном таком аспекте указанный способ дополнительно включает введение субъекту эффективного количества по меньшей мере одного дополнительного терапевтического агента, как описано ниже. «Субъект» в соответствии с любым из вышеприведенных аспектов может быть человеком.
В дополнительном аспекте в данном документе предложены фармацевтические составы, содержащие любую из предложенных в данном документе биспецифических агонистических CD28-антигенсвязывающих молекул, например, для применения в любом из вышеприведенных терапевтических способов. В одном аспекте фармацевтический состав содержит любую из предложенных в данном документе биспецифических агонистических CD28-антигенсвязывающих молекул и фармацевтически приемлемый носитель. В другом аспекте фармацевтический состав содержит любую из предложенных в данном документе биспецифических агонистических CD28-антигенсвязывающих молекул и по меньшей мере один дополнительный терапевтический агент.
Предложенные в данном документе биспецифические агонистические CD28-антигенсвязывающие молекулы можно применять отдельно или в комбинации с другими агентами в терапии. Например, предложенные в данном документе биспецифические агонистические CD28-антигенсвязывающие молекулы можно вводить совместно с по меньшей мере одним дополнительным терапевтическим агентом.
Такие комбинированные виды терапии, упомянутые выше, охватывают комбинированное введение (в котором два или более терапевтических агентов включены в один или в отдельные составы), а также раздельное введение, в случае чего введение предложенного в данном документе антитела может происходить до, одновременно и/или после введения дополнительного терапевтического агента или агентов. В одном аспекте введение биспецифической агонистической CD28-антигенсвязывающей молекулы и введение дополнительного терапевтического агента может разделять около одного месяца, или около одной, двух или трех недель, или около одних, двух, трех, четырех, пяти или шести суток.
Предложенную в данном документе антигенсвязывающую молекулу (и любой дополнительный терапевтический агент) можно вводить любым подходящим способом, включая парентеральное, внутрилегочное и интраназальное, и, если необходимо для местного лечения, внутриочаговое введение. Парентеральные инфузии включают внутримышечное, внутривенное, внутриартериальное, внутрибрюшинное или подкожное введение. Введение дозы можно проводить любым удобным способом, например путем инъекций, таких как внутривенные или подкожные инъекции, частично в зависимости от того, является ли введение кратковременным или постоянным. В данном документе предусмотрены различные схемы дозирования, включая, но не ограничиваясь этим, однократное или многократное введение в различные моменты времени, болюсное введение и импульсную инфузию.
Описанные в данном документе биспецифические агонистические CD28-антигенсвязывающие молекулы необходимо составлять, дозировать и вводить способом, соответствующим требованиям надлежащей медицинской практики. Факторы, которые необходимо учитывать в этом контексте, включают конкретное нарушение, подлежащее лечению, конкретное млекопитающее, подлежащее лечению, клиническое состояние отдельного пациента, причину нарушения, место доставки агента, способ введения, график введения и другие факторы, известные практикующим врачам. Биспецифическую агонистическую СD28-антигенсвязывающую молекулу необязательно составляют с одним или более агентами, применяемыми в настоящее время для предотвращения или лечения рассматриваемого нарушения. Эффективное количество таких других агентов зависит от количества антитела, присутствующего в составе, типа нарушения или лечения и других факторов, которые обсуждались выше. Другие агенты обычно применяют в таких же дозировках и вводят путями, описанными в данном документе, или в дозировках, составляющих от около 1 до 99% дозировок, описанных в данном документе, или в любой дозировке и любым путем, которые эмпирически/клинически определены как подходящие.
Для профилактики или лечения заболевания соответствующая доза описанной в данном документе биспецифической агонистической CD28-антигенсвязывающей молекулы (применяемой отдельно или в комбинации с одним или более другими дополнительными терапевтическими агентами) зависит от типа заболевания, подлежащего лечению, типа антитела, тяжести и течения заболевания, того, вводится ли антитело с профилактической или терапевтической целью, предыдущей терапии, истории болезни пациента и его ответа на антитела, а также решения лечащего врача. Биспецифическую агонистическую СD28-антигенсвязывающую молекулу вводят пациенту подходящим образом за один раз или в течение серии процедур. В зависимости от типа и тяжести заболевания, начальная кандидатная дозировка биспецифической агонистической СD28-антигенсвязывающей молекулы для введения пациенту может составлять от около 1 мкг/кг до 15 мг/кг (например, 0,5 мг/кг - 10 мг/кг), независимо от того, например, осуществляют ли одно или более отдельных введений или непрерывную инфузию. Одна типичная суточная доза может варьироваться от около 1 мкг/кг до 100 мг/кг или более, в зависимости от факторов, упомянутых выше. В случае повторных введений в течение нескольких суток или более, в зависимости от состояния, лечение в общем случае проводят до достижения необходимой степени подавления симптомов заболевания. Одна типовая дозировка антитела находится в диапазоне от около 0,05 мг/кг до около 10 мг/кг. Таким образом, пациенту можно вводить одну или более доз, составляющих около 0,5 мг/кг, 2,0 мг/кг, 4,0 мг/кг или 10 мг/кг (или любую их комбинацию). Такие дозы можно вводить с интервалами, например, раз в неделю или раз в три недели (например, чтобы пациент получал от около двух до около двадцати или, например, около шести доз антитела). Можно вводить начальную более высокую нагрузочную дозу, за которой следует одна или более меньших доз. При этом можно применять другие схемы дозирования. Эффективность такой терапии легко контролировать при помощи традиционных методик и анализов.
Другие агенты и варианты лечения
Биспецифические агонистические СD28-антигенсвязывающие молекулы по изобретению можно вводить в комбинации с одним или более агентами в терапии. Например, антигенсвязывающую молекулу по изобретению можно вводить совместно с по меньшей мере одним дополнительным терапевтическим агентом. Термин «терапевтический агент» охватывает любой агент, который можно вводить для лечения симптома или заболевания у индивида, нуждающегося в таком лечении. Такой дополнительный терапевтический агент может содержать любые активные ингредиенты, подходящие для конкретного показания, подлежащего лечению, предпочтительно те, которые обладают дополняющими видами активности и не оказывают отрицательного влияния друг на друга. В определенных вариантах осуществления дополнительный терапевтический агент представляет собой другой противораковый агент, например, разрушитель микротрубочек, антиметаболит, ингибитор топоизомеразы, интеркалятор ДНК, алкилирующий агент, гормональную терапию, ингибитор киназы, антагонист рецептора, активатор апоптоза опухолевых клеток или антиангиогенный агент. В определенных аспектах дополнительный терапевтический агент представляет собой иммуномодулирующий агент, цитостатический агент, ингибитор клеточной адгезии, цитотоксический или цитостатический агент, активатор клеточного апоптоза или агент, который повышает чувствительность клеток к индукторам апоптоза.
Таким образом, предложены биспецифические агонистические CD28-антигенсвязывающие молекулы по изобретению или содержащие их фармацевтические композиции для применения в лечении рака, причем биспецифическую антигенсвязывающую молекулу вводят в комбинации с химиотерапевтическим агентом, лучевой терапией и/или другими агентами для применения в иммунотерапии рака.
Такие другие агенты приемлемо присутствуют в комбинации в количествах, которые эффективны для предполагаемой цели. Эффективное количество таких других агентов зависит от количества используемого слитого белка, типа нарушения или лечения и других факторов, обсуждаемых выше. Биспецифическую антигенсвязывающую молекулу или антитело по изобретению в общем случае применяют в таких же дозировках и вводят такими же путями, как описано в данном документе, или в дозировках, составляющих от около 1 до 99% дозировок, описанных в данном документе, или в любой дозировке и любым путем, которые эмпирически/клинически определены как подходящие. Такие виды комбинированной терапии, отмеченные выше, охватывают комбинированное введение (при котором два или более терапевтических агентов включены в одну или в отдельные композиции) и раздельное введение, в случае которого введение биспецифической антигенсвязывающей молекулы или антитела по изобретению может происходить до, одновременно и/или после введения дополнительного терапевтического агента и/или адъюванта.
В дополнительном аспекте предложена биспецифическая агонистическая СD28-антигенсвязывающая молекула, описанная в данном документе выше, для применения в лечении рака, причем биспецифическую антигенсвязывающую молекулу вводят в комбинации с другим иммуномодулятором. Термин «иммуномодулятор» относится к любому веществу, включая моноклональное антитело, которое оказывает воздействие на иммунную систему. Молекулы по изобретению можно считать иммуномодуляторами. Иммуномодуляторы можно использовать в качестве антинеопластических агентов для лечения рака. В одном аспекте иммуномодуляторы включают, но не ограничиваются этим, анти-CTLA4 антитела (например, ипилимумаб), анти-PD1 антитела (например, ниволумаб или пембролизумаб), антитела к PD-L1 (например, атезолизумаб, авелумаб или дурвалумаб), антитела к ОХ-40, антитела к 4-1ВВ и антитела к GITR. Такие виды комбинированной терапии, отмеченные выше, охватывают комбинированное введение (при котором два или более терапевтических агентов включены в одну или в отдельные композиции) и раздельное введение, в случае которого введение биспецифической антигенсвязывающей молекулы может происходить до, одновременно и/или после введения дополнительного терапевтического агента и/или адъюванта.
Комбинация с Т-клеточными биспецифическими антителами
В одном аспекте биспецифические агонистические CD28-антигенсвязывающие молекулы по изобретению можно вводить в комбинации с активирующими Т-клетки анти-СD3 биспецифическими антителами. В одном аспекте активирующее Т-клетки анти-СD3 биспецифическое антитело, специфическое в отношении опухолеассоциированного антигена, представляет собой анти-СЕА/анти-СD3 биспецифическое антитело или анти-MCSP/анти-СD3 биспецифическое антитело. В одном конкретном аспекте активирующее Т-клетки анти-СD3 биспецифическое антитело, специфическое в отношении опухолеассоциированного антигена, представляет собой анти-СЕА/анти-СD3 биспецифическое антитело.
В одном аспекте биспецифическая агонистическая CD28-антигенсвязывающая молекула содержит по меньшей мере один антигенсвязывающий домен, способный к специфическому связыванию с опухолеассоциированным антигеном (ОАА), выбранным из группы, состоящей из белка активации фибробластов (FAP), карциноэмбрионального антигена (СЕА), рецептора фолиевой кислоты альфа (FolR1), ассоциированного с меланомой хондроитинсульфата протеогликана (MCSP), рецептора эпидермального фактора роста (EGFR), рецептора 2 эпидермального фактора роста человека (HER2) и p95HER2, подходит для введения в комбинации с анти-CEA/анти-CD3 биспецифическим антителом. В другом конкретном аспекте ОАА выбран из группы, состоящей из белка активации фибробластов (FAP), карциноэмбрионального антигена (СЕА), ЕрСАМ, HER3, CD30 или TPBG (5Т4).
В конкретном аспекте анти-СD3 биспецифическое антитело для применения в комбинации содержит первый антигенсвязывающий домен, содержащий вариабельную область тяжелой цепи (VHCD3), содержащую последовательность CDR-H1 SEQ ID NO: 439, последовательность CDR-H2 SEQ ID NO: 440 и последовательность CDR-H3 SEQ ID NO: 441; и/или вариабельную область легкой цепи (VLCD3), содержащую последовательность CDR-L1 SEQ ID NO: 442, последовательность CDR-L2 SEQ ID NO: 443 и последовательность CDR-L3 SEQ ID NO: 444. В частности, анти-СD3 биспецифическое антитело содержит первый антигенсвязывающий домен, содержащий вариабельную область тяжелой цепи (VHCD3), которая является по меньшей мере на 90%, 95%, 96%, 97%, 98% или 99% идентичной аминокислотной последовательности SEQ ID NO: 445, и вариабельную область легкой цепи (VLCD3), которая является по меньшей мере на около 90%, 95%, 96%, 97%,
98% или 99% идентичной аминокислотной последовательности SEQ ID NO: 446. В дополнительном аспекте анти-СD3 биспецифическое антитело содержит вариабельную область тяжелой цепи (VHCD3), содержащую аминокислотную последовательность SEQ ID NO: 445, и/или вариабельную область легкой цепи (VLCD3), содержащую аминокислотную последовательность SEQ ID NO: 446.
В другом аспекте анти-СD3 биспецифическое антитело для применения в комбинации содержит первый антигенсвязывающий домен, содержащий вариабельную область тяжелой цепи (VHCD3), содержащую последовательность CDR-H1 SEQ ID NO: 596, последовательность CDR-H2 SEQ ID NO: 597 и последовательность CDR-H3 SEQ ID NO: 598; и/или вариабельную область легкой цепи (VLCD3), содержащую последовательность CDR-L1 SEQ ID NO: 599, последовательность CDR-L2 SEQ ID NO: 600 и последовательность CDR-L3 SEQ ID NO: 601. В частности, анти-СD3 биспецифическое антитело содержит первый антигенсвязывающий домен, содержащий вариабельную область тяжелой цепи (VHCD3), которая является по меньшей мере на 90%, 95%, 96%, 97%, 98% или 99% идентичной аминокислотной последовательности SEQ ID NO: 602, и вариабельную область легкой цепи (VLCD3), которая является по меньшей мере на около 90%, 95%, 96%, 97%, 98% или 99% идентичной аминокислотной последовательности SEQ ID NO: 603. В дополнительном аспекте анти-CD20/анти-CD3 биспецифическое антитело содержит вариабельную область тяжелой цепи (VHCD3), содержащую аминокислотную последовательность SEQ ID NO: 602, и/или вариабельную область легкой цепи (VLCD3), содержащую аминокислотную последовательность SEQ ID NO: 603.
В другом аспекте анти-СD3 биспецифическое антитело для применения в комбинации содержит первый антигенсвязывающий домен, содержащий вариабельную область тяжелой цепи (VHCD3), содержащую последовательность CDR-H1 SEQ ID NO: 604, последовательность CDR-H2 SEQ ID NO: 605 и последовательность CDR-H3 SEQ ID NO: 606; и/или вариабельную область легкой цепи (VLCD3), содержащую последовательность CDR-L1 SEQ ID NO: 607, последовательность CDR-L2 SEQ ID NO: 608 и последовательность CDR-L3 SEQ ID NO: 609. В частности, анти-СD3 биспецифическое антитело содержит первый антигенсвязывающий домен, содержащий вариабельную область тяжелой цепи (VHCD3), которая является по меньшей мере на 90%, 95%, 96%, 97%, 98% или 99% идентичной аминокислотной последовательности SEQ ID NO: 610, и вариабельную область легкой цепи (VLCD3), которая является по меньшей мере на около 90%, 95%, 96%, 97%, 98% или 99% идентичной аминокислотной последовательности SEQ ID NO: 611. В дополнительном аспекте анти-CD20/анти-CD3 биспецифическое антитело содержит вариабельную область тяжелой цепи (VHCD3), содержащую аминокислотную последовательность SEQ ID NO: 610, и/или вариабельную область легкой цепи (VLCD3), содержащую аминокислотную последовательность SEQ ID NO: 611.
В одном конкретном аспекте анти-СЕА/анти-СD3 биспецифическое антитело содержит полипептид, который является по меньшей мере на 95%, 96%, 97%, 98% или 99% идентичным последовательности SEQ ID NO: 161, полипептид, который является по меньшей мере на 95%, 96%, 97%, 98% или 99% идентичным последовательности SEQ ID NO: 162, полипептид, который является по меньшей мере на 95%, 96%, 97%, 98% или 99% идентичным последовательности SEQ ID NO: 163, и полипептид, который является по меньшей мере на 95%, 96%, 97%, 98% или 99% идентичным последовательности SEQ ID NO: 164. В дополнительном конкретном варианте осуществления биспецифическое антитело содержит полипептидную последовательность SEQ ID NO: 161, полипептидную последовательность SEQ ID NO: 162, полипептидную последовательность SEQ ID NO: 163 и полипептидную последовательность SEQ ID NO: 164 (CEA CD3 TCB).
В другом конкретном аспекте анти-СЕА/анти-СD3 биспецифическое антитело содержит полипептид, который является по меньшей мере на 95%, 96%, 97%, 98% или 99% идентичным последовательности SEQ ID NO: 165, полипептид, который является по меньшей мере на 95%, 96%, 97%, 98% или 99% идентичным последовательности SEQ ID NO: 166, полипептид, который является по меньшей мере на 95%, 96%, 97%, 98% или 99% идентичным последовательности SEQ ID NO: 167, и полипептид, который является по меньшей мере на 95%, 96%, 97%, 98% или 99% идентичным последовательности SEQ ID NO: 168. В дополнительном конкретном варианте осуществления биспецифическое антитело содержит полипептидную последовательность SEQ ID NO: 165, полипептидную последовательность SEQ ID NO: 166, полипептидную последовательность SEQ ID NO: 167 и полипептидную последовательность SEQ ID NO: 168 (СЕАСАМ5 CD3 TCB).
Конкретные биспецифические антитела дополнительно описаны в публикации РСТ № WO2014/131712 А1. В дополнительном аспекте анти-СЕА/анти-СD3 биспецифическое антитело также может содержать привлекающий Т-клетки биспецифический активатор (BiTE®). В дополнительном аспекте анти-СЕА/анти-СD3 биспецифическое антитело представляет собой биспецифическое антитело, описанное в WO2007/071426 или WO2014/131712.
В другом аспекте биспецифическую агонистическую CD28-антигенсвязывающую молекулу по изобретению, содержащую антигенсвязывающий домен, способный к специфическому связыванию с поверхностным антигеном В-клеток, можно вводить в комбинации с активирующими Т-клетки анти-СD3 биспецифическими антителами. В одном аспекте активирующее Т-клетки анти-СD3 биспецифическое антитело является специфическим в отношении поверхностного антигена В-клеток, в частности представляет собой анти-СD20/анти-СD3 биспецифическое антитело.
В контексте данного документа анти-СD20/анти-СD3 биспецифические антитела представляют собой биспецифические антитела, содержащие первый антигенсвязывающий домен, который связывается с CD3, и второй антигенсвязывающий домен, который связывается с CD20. Таким образом, в контексте данного документа анти-СD20/анти-СD3 биспецифическое антитело содержит первый антигенсвязывающий домен, содержащий вариабельную область тяжелой цепи (VHCD3) и вариабельную область легкой цепи (VLCD3), и второй антигенсвязывающий домен, содержащий вариабельную область тяжелой цепи (VHCD20) и вариабельную область легкой цепи (VLCD20).
В конкретном аспекте анти-СD20/анти-СD3 биспецифическое антитело для применения в комбинации содержит первый антигенсвязывающий домен, содержащий вариабельную область тяжелой цепи (VHCD3), содержащую последовательность CDR-H1 SEQ ID NO: 439, последовательность CDR-H2 SEQ ID NO: 440 и последовательность CDR-H3 SEQ ID NO: 441; и/или вариабельную область легкой цепи (VLCD3), содержащую последовательность CDR-L1 SEQ ID NO: 442, последовательность CDR-L2 SEQ ID NO: 443 и последовательность CDR-L3 SEQ ID NO: 444. В частности, анти-СD20/анти-СD3 биспецифическое антитело содержит первый антигенсвязывающий домен, содержащий вариабельную область тяжелой цепи (VHCD3), которая является по меньшей мере на 90%, 95%, 96%, 97%, 98% или 99% идентичной аминокислотной последовательности SEQ ID NO: 445, и вариабельную область легкой цепи (VLCD3), которая является по меньшей мере на около 90%, 95%, 96%, 97%, 98% или 99% идентичной аминокислотной последовательности SEQ ID NO: 446. В дополнительном аспекте анти-СD20/анти-СD3 биспецифическое антитело содержит вариабельную область тяжелой цепи (VHCD3), содержащую аминокислотную последовательность SEQ ID NO: 445, и/или вариабельную область легкой цепи (VLCD3), содержащую аминокислотную последовательность SEQ ID NO: 446.
В одном аспекте антитело, которое специфически связывается с CD3, представляет собой полноразмерное антитело. В одном аспекте антитело, которое специфически связывается с CD3, представляет собой антитело класса IgG человека, в частности антитело класса IgG1 человека. В одном аспекте антитело, которое специфически связывается с CD3, представляет собой фрагмент антитела, в частности молекулу Fab или молекулу scFv, конкретнее - молекулу Fab. В одном аспекте антитело, которое специфически связывается с CD3, представляет собой кроссоверную молекулу Fab, в которое обменены (т.е. замещены друг другом) вариабельные домены или константные домены тяжелой и легкой цепи Fab. В одном аспекте антитело, которое специфически связывается с CD3, представляет собой гуманизированное антитело.
В другом аспекте анти-СD20/анти-СD3 биспецифическое антитело содержит второй антигенсвязывающий домен, содержащий вариабельную область тяжелой цепи (VHCD20), содержащую последовательность CDR-H1 SEQ ID NO: 447, последовательность CDR-H2 SEQ ID NO: 448 и последовательность CDR-H3 SEQ ID NO: 449, и/или вариабельную область легкой цепи (VLCD20), содержащую последовательность CDR-L1 SEQ ID NO: 450, последовательность CDR-L2 SEQ ID NO: 451 и последовательность CDR-L3 SEQ ID NO: 452. В частности, анти-СD20/анти-СD3 биспецифическое антитело содержит второй антигенсвязывающий домен, содержащий вариабельную область тяжелой цепи (VHCD20), которая является по меньшей мере на 90%, 95%, 96%, 97%, 98% или 99% идентичной аминокислотной последовательности SEQ ID NO: 453, и вариабельную область легкой цепи (VLCD20), которая является по меньшей мере на около 90%, 95%, 96%, 97%, 98% или 99% идентичной аминокислотной последовательности SEQ ID NO: 454. В дополнительном аспекте анти-CD20/анти-CD3 биспецифическое антитело содержит второй антигенсвязывающий домен, содержащий вариабельную область тяжелой цепи (VHCD20), содержащую аминокислотную последовательность SEQ ID NO: 453, и/или вариабельную область легкой цепи (VLCD20), содержащую аминокислотную последовательность SEQ ID NO: 454.
В другом конкретном аспекте анти-СD20/анти-СD3 биспецифическое антитело содержит третий антигенсвязывающий домен, который связывается с CD20. В частности, анти-СD20/анти-СD3 биспецифическое антитело содержит третий антигенсвязывающий домен, содержащий вариабельную область тяжелой цепи (VHCD20), содержащую последовательность CDR-H1 SEQ ID NO: 447, последовательность CDR-H2 SEQ ID NO: 448 и последовательность CDR-H3 SEQ ID NO: 449, и/или вариабельную область легкой цепи (VLCD20), содержащую последовательность CDR-L1 SEQ ID NO: 450, последовательность CDR-L2 SEQ ID NO: 451 и последовательность CDR-L3 SEQ ID NO: 452. Конкретнее, анти-CD20/анти-CD3 биспецифическое антитело содержит третий антигенсвязывающий домен, содержащий вариабельную область тяжелой цепи (VHCD20), которая является по меньшей мере на 90%, 95%, 96%, 97%, 98% или 99% идентичной аминокислотной последовательности SEQ ID NO: 453, и вариабельную область легкой цепи (VLCD20), которая является по меньшей мере на около 90%, 95%, 96%, 97%, 98% или 99% идентичной аминокислотной последовательности SEQ ID NO: 454. В дополнительном аспекте анти-CD20/анти-CD3 биспецифическое антитело содержит третий антигенсвязывающий домен, содержащий вариабельную область тяжелой цепи (VHCD20), содержащую аминокислотную последовательность SEQ ID NO: 453, и/или вариабельную область легкой цепи (VLCD20), содержащую аминокислотную последовательность SEQ ID NO: 454.
В дополнительном аспекте анти-СD20/анти-СD3 биспецифическое антитело представляет собой биспецифическое антитело, в котором первый антигенсвязывающий домен представляет собой молекулу кросс-Fab, в которой обменены вариабельные домены или константные домены тяжелой и легкой цепи Fab, а второй и, в случае наличия, третий антигенсвязывающий домен представляет собой стандартную молекулу Fab.
В другом аспекте анти-СD20/анти-СD3 биспецифическое антитело представляет собой биспецифическое антитело, в котором (i) второй антигенсвязывающий домен слит на С-конце тяжелой цепи Fab с N-концом тяжелой цепи Fab первого антигенсвязывающего домена, первый антигенсвязывающий домен слит на С-конце тяжелой цепи Fab с N-концом первой субъединицы Fc-домена, а третий антигенсвязывающий домен слит на С-конце тяжелой цепи Fab с N-концом второй субъединицы Fc-домена, или (ii) первый антигенсвязывающий домен слит на С-конце тяжелой цепи Fab с N-концом тяжелой цепи Fab второго антигенсвязывающего домена, второй антигенсвязывающий домен слит на С-конце тяжелой цепи Fab с N-концом первой субъединицы Fc-домена, а третий антигенсвязывающий домен слит на С-конце тяжелой цепи Fab с N-концом второй субъединицы Fc-домена.
Молекулы Fab могут быть слиты с Fc-доменом или друг с другом напрямую или посредством пептидного линкера, содержащего одну или более аминокислот, как правило, около 2-20 аминокислот. Пептидные линкеры известны в данной области техники и описаны в данном документе. В одном аспекте пептидный линкер представляет собой (G4S)2 (SEQ ID NO: 147). Другой такой подходящий линкер содержит последовательность (G4S)4 (SEQ ID NO: 152). Дополнительно, линкеры могут содержать шарнирную область иммуноглобулина (или ее часть). В частности, если молекула Fab с N-концом субъединицы Fc-домена, она может быть слита посредством шарнирной области иммуноглобулина или ее части с дополнительным пептидным линкером или без него. В дополнительном аспекте анти-СD20/анти-СD3 биспецифическое антитело содержит Fc-домен, содержащий одну или более аминокислотных замен, которые снижают связывание Fc-рецептора и/или эффекторную функцию. В частности анти-СD20/анти-СD3 биспецифическое антитело содержит Fc-домен IgG1, содержащий аминокислотные замены L234A, L235A и P329G.
В конкретном аспекте анти-СD20/анти-СD3 биспецифическое антитело содержит полипептид, который является по меньшей мере на 95%, 96%, 97%, 98% или 99% идентичным последовательности SEQ ID NO: 455, полипептид, который является по меньшей мере на 95%, 96%, 97%, 98% или 99% идентичным последовательности SEQ ID NO: 456, полипептид, который является по меньшей мере на 95%, 96%, 97%, 98% или 99% идентичным последовательности SEQ ID NO: 457, и полипептид, который является по меньшей мере на 95%, 96%, 97%, 98% или 99% идентичным последовательности SEQ ID NO: 458. В дополнительном конкретном варианте осуществления биспецифическое антитело содержит полипептидную последовательность SEQ ID NO: 455, полипептидную последовательность SEQ ID NO: 456, полипептидную последовательность SEQ ID NO: 457 и полипептидную последовательность SEQ ID NO: 458 (CD20 ТСВ).
Конкретные биспецифические антитела описаны в публикации РСТ № WO2016/020309 А1 или в WO2015/095392 А1. В дополнительном аспекте анти-CD20/анти-CD3 биспецифическое антитело также может содержать привлекающий Т-клетки биспецифический активатор (BiTE®). В дополнительном аспекте анти-СD20/анти-СD3 биспецифическое антитело представляет собой XmAb® 13676. В другом аспекте биспецифическое антитело представляет собой REGN1979. В другом аспекте биспецифическое антитело представляет собой FBTA05 (Lymphomun).
В другом аспекте биспецифическая агонистическая CD28-антигенсвязывающая молекула по изобретению предназначена для применения в способе лечения или замедления прогрессирования рака, при этом биспецифическую агонистическую СD28-антигенсвязывающую молекулу применяют в комбинации с нти-СО20/анти-СD3 биспецифическим антителом, и дополнительно их комбинируют с агентом, блокирующим взаимодействие PD-L1/PD-1. Агент, блокирующий взаимодействие с PD-L1/PD-1, является антагонистом связывания PD-L1 или антагонистом связывания PD-1. В частности, агент, блокирующий взаимодействие PD-L1/PD-1, представляет собой анти-PD-L1 антитело или анти-PD-1 антитело.
В другом аспекте биспецифическую агонистическую CD28-антигенсвязывающую молекулу по изобретению, содержащую антигенсвязывающий домен, способный к специфическому связыванию с антигеном клеточной поверхности ММ, можно вводить в комбинации с активирующими Т-клетки анти-СD3 биспецифическими антителами. В одном аспекте активирующее Т-клетки анти-СD3 биспецифическое антитело является специфическим в отношении антигена клеточной поверхности ММ, в частности представляет собой анти-GPRC5D/анти-CD3 биспецифическое антитело.
В одном конкретном аспекте анти-GPRC5D/анти-CD3 биспецифическое антитело содержит полипептид, который является по меньшей мере на 95%, 96%, 97%, 98% или 99% идентичным последовательности SEQ ID NO: 398, полипептид, который является по меньшей мере на 95%, 96%, 97%, 98% или 99% идентичным последовательности SEQ ID NO: 399, полипептид, который является по меньшей мере на 95%, 96%, 97%, 98% или 99% идентичным последовательности SEQ ID NO: 404, и полипептид, который является по меньшей мере на 95%, 96%, 97%, 98% или 99% идентичным последовательности SEQ ID NO: 405. В дополнительном конкретном аспекте биспецифическое антитело содержит полипептидную последовательность SEQ ID NO: 398, полипептидную последовательность SEQ ID NO: 399, полипептидную последовательность SEQ ID NO: 404 и полипептидную последовательность SEQ ID NO: 405 (GPRC5D CD3 ТСВ).
В другом аспекте предложен комбинированный продукт, содержащий описанную в данном документе биспецифическую агонистическую CD28-антигенсвязывающую молекулу и активирующее Т-клетки анти-СD3 биспецифическое антитело. В одном аспекте активирующее Т-клетки анти-СD3 биспецифическое антитело, специфическое в отношении опухолеассоциированного антигена, представляет собой анти-СЕА/анти-СD3 биспецифическое антитело или анти-МСSР/анти-СD3 биспецифическое антитело. В одном конкретном аспекте активирующее Т-клетки анти-СD3 биспецифическое антитело, специфическое в отношении опухолеассоциированного антигена, представляет собой анти-СЕА/анти-СD3 биспецифическое антитело. В другом аспекте активирующее Т-клетки анти-СD3 биспецифическое антитело, специфическое в отношении опухолеассоциированного антигена, представляет собой анти-СD20/анти-СD3 биспецифическое антитело. В дополнительном аспекте активирующее Т-клетки анти-СD3 биспецифическое антитело, специфическое в отношении опухолеассоциированного антигена, представляет собой анти-GPRC5D/анти-СD3 биспецифическое антитело.
Комбинация с агентами, блокирующими взаимодействие PD-L1/PD-1
В одном аспекте биспецифические агонистические CD28-антигенсвязывающие молекулы по изобретению можно вводить в комбинации с агентами, блокирующими взаимодействие PD-L1/PD-1, такими как антагонист связывания PD-L1 или антагонист связывания PD-1, в частности анти-PD-L1 антитело или анти-PD-1 антитело.
В одном аспекте агент, блокирующий взаимодействие PD-L1/PD-1, представляет собой анти-PD-L1 антитело. Термин «PD-L1», также известный как CD274 или В7-Н1, относится к любому нативному PD-L1 из любого источника, относящегося к позвоночным, включая млекопитающих, таких как приматы (например, люди), отличные от человека приматы (например, яванские макаки) и грызуны (например, мыши и крысы), в частности к «PD-L1 человека». Аминокислотная последовательность полного PD-L1 человека приведена под номером доступа UniProt (www.uniprot.org) № Q9NZQ7 (SEQ ID NO: 459). Термин «антагонист связывания PD-L1» относится к молекуле, которая снижает, блокирует, ингибирует, подавляет или препятствует передаче сигнала в результате взаимодействия PD-L1 с одним или более его партнерами по связыванию, такими как PD-1, В7-1. В некоторых аспектах антагонист связывания PD-L1 представляет собой молекулу, которая ингибирует связывание PD-L1 с его партнерами по связыванию. В конкретном аспекте PD-L1-связывающий антагонист ингибирует связывание PD-L1 с PD-1 и/или В7-1. В некоторых аспектах антагонисты связывания PD-L1 включают антитела против PD-L1, их антигенсвязывающие фрагменты, иммуноадгезины, слитые белки, олигопептиды и другие молекулы, которые снижают, блокируют, ингибируют, подавляют или препятствует передаче сигнала в результате взаимодействия PD-L1 с одним или более его партнерами по связыванию, такими как PD-1, В7-1. В одном аспекте антагонист связывания PD-L1 снижает негативный костимулирующий сигнал, опосредованный или передаваемый клеточными поверхностными белками, экспрессируемыми на Т-лимфоцитах, которые опосредуют сигнализацию через PD-L1, так, чтобы сделать дисфункциональную Т-клетку менее дисфункциональной (например, повышая эффекторные ответы на распознавание антигена). В частности, антагонист связывания PD-L1 представляет собой анти-PD-L1 антитело. Термины «анти-PD-L1 антитело», или «антитело, связывающееся с PD-L1 человека», или «антитело, которое специфически связывается с PD-L1 человека», или «антагонистическое анти-PD-L1» относятся к антителу, которое специфически связывается с PD-L1 человека с аффинностью связывания со значением KD 1,0×10-8 моль/л или меньше, в одном аспекте - со значением 1,0×10-9 моль/л или меньше. Аффинность связывания определяют с помощью стандартного анализа связывания, такого как метод поверхностного плазмонного резонанса (BIAcore®, GE-Healthcare Uppsala, Sweden). В конкретном аспекте агент, блокирующий взаимодействие PD-L1/PD-1, представляет собой анти-PD-L1 антитело. В конкретном аспекте анти-PD-L1 антитело выбрано из группы, состоящей из атезолизумаба (MPDL3280A, RG7446), дурвалумаба (MEDI4736), авелумаба (MSB0010718C) и MDX-1105. В конкретном аспекте анти-PD-L1 антитело представляет собой YW243.55.S70, описанное в данном документе. В другом конкретном аспекте анти-PD-L1 антитело представляет собой MDX-1105, описанное в данном документе. В другом конкретном аспекте анти-PD-L1 антитело представляет собой MEDI4736 (дурвалумаб). В дополнительном аспекте анти-PD-L1 антитело представляет собой MSB0010718C (авелумаб). в частности, агент, блокирующий взаимодействие PD-L1/PD-1, представляет собой атезолизумаб (MPDL3280A). В другом аспекте агент, блокирующий взаимодействие PD-L1/PD-1, представляет собой анти-PD-L1 антитело, содержащее вариабельный домен тяжелой цепи VH(PDL-1) с SEQ ID NO: 460 и вариабельный домен легкой цепи VL(PDL-1) с SEQ ID NO: 461. В другом аспекте агент, блокирующий взаимодействие PD-L1/PD-1, представляет собой анти-PD-L1 антитело, содержащее вариабельный домен тяжелой цепи VH(PDL-1) с SEQ ID NO: 462 и вариабельный домен легкой цепи VL(PDL-1) с SEQ ID NO: 463.
Термин «PD-1», также известный как CD279, PD1 или белок запрограммированной гибели клеток 1, относится к нативному PD-L1 из любого источника, относящегося к позвоночным, включая млекопитающих, таких как приматы (например, люди), отличные от человека приматы (например, яванские макаки) и грызуны (например, мыши и крысы), в частности к белку PD-1 человека с аминокислотной последовательностью, приведенной по номером доступа UniProt (www.uniprot.org) № Q15116 (SEQ ID NO: 464). Термин «антагонист связывания PD-1» относится к молекуле, которая ингибирует связывание PD-1 с его лигандами партнерами по связыванию. В некоторых вариантах осуществления антагонист связывания PD-1 ингибирует связывание PD-1 с PD-L1. В некоторых вариантах осуществления антагонист связывания PD-1 ингибирует связывание PD-1 с PD-L2. В некоторых вариантах осуществления антагонист связывания PD-1 ингибирует связывание PD-1 как с PD-L1, так и с PD-L2. В частности, антагонист связывания PD-L1 представляет собой анти-PD-L1 антитело. Термины «анти-PD-1 антитело», или «антитело, связывающееся с PD-1 человека», или «антитело, которое специфически связывается с PD-1 человека», или «антагонистическое анти-PD-1» относятся к антителу, которое специфически связывается с PD1 человека с аффинностью связывания со значением KD 1,0×10-8 моль/л или меньше, в одном аспекте - со значением KD 1,0×10-9 моль/л или меньше. Аффинность связывания определяют с помощью стандартного анализа связывания, такого как метод поверхностного плазмонного резонанса (BIAcore®, GE-Healthcare Uppsala, Sweden). В одном аспекте агент, блокирующий взаимодействие PD-L1/PD-1, представляет собой анти-PD-1 антитело. В конкретном аспекте анти-PD-1 антитело выбрано из группы, состоящей из MDX 1106 (ниволумаба), МK-3475 (пембролизумаба), СТ-011 (пидилизумаба), MEDI-0680 (АМР-514), PDR001, REGN2810 и BGB-108, в частности из пембролизумаба и ниволумаба. В другом аспекте агент, блокирующий взаимодействие PD-L1/PD-1, представляет собой анти-PD-1 антитело, содержащее вариабельный домен тяжелой цепи VH(PD-1) с SEQ ID NO: 465 и вариабельный домен легкой цепи VL(PD-1) с SEQ ID NO: 466. В другом аспекте агент, блокирующий взаимодействие PD-L1/PD-1, представляет собой анти-PD-1 антитело, содержащее вариабельный домен тяжелой цепи VH(PD-1) с SEQ ID NO: 467 и вариабельный домен легкой цепи VL(PD-1) с SEQ ID NO: 468.
В другом аспекте предложен комбинированный продукт, содержащий описанную в данном документе биспецифическую агонистическую CD28-антигенсвязывающую молекулу и агенты, блокирующие взаимодействие PD-L1/PD-1, такие как антагонист связывания PD-L1 или антагонист связывания PD-1, в частности анти-PD-L1 антитело или анти-PD-1 антитело.
Такие комбинированные виды терапии, упомянутые выше, охватывают комбинированное введение (в котором два или более терапевтических агентов включены в один или в отдельные составы), а также раздельное введение, в случае чего введение терапевтического агента может происходить до, одновременно и/или после введения дополнительного терапевтического агента или агентов. В одном варианте осуществления введение терапевтического агента и введение дополнительного терапевтического агента может разделять около одного месяца, или около одной, двух или трех недель, или около одних, двух, трех, четырех, пяти или шести суток. Готовые изделия
В другом аспекте данного изобретения предложено готовое изделие, содержащее материалы, применяемые при лечении, предотвращении и/или диагностике вышеописанных нарушений. Готовое изделие содержит контейнер и этикетку или вкладыш в упаковку на нем или связанные с ним. Подходящие контейнеры включают, например, бутылки, флаконы, шприцы, пакеты для в/в раствора и т.д. Контейнеры могут быть выполнены из ряда материалов, таких как стекло или пластик. Контейнер содержит композицию, которая сама по себе или в комбинации с другой композицией эффективна для лечения, предотвращения и/или диагностики патологического состояния, и может иметь стерильное входное отверстие (например, контейнер может представлять собой пакет для внутривенного раствора или флакон с пробкой, прокалываемой гиподермической иглой для инъекции). По меньшей мере один активный агент в композиции представляет собой биспецифическую агонистическую CD28-антигенсвязывающую молекулу по изобретению. На этикетке или на вкладыше в упаковку указано, что композицию применяют для лечения выбранного патологического состояния. Кроме того, готовое изделие может содержать (а) первый контейнер с содержащейся в нем композицией, причем указанная композиция содержит биспецифическую агонистическую CD28-антигенсвязывающую молекулу по изобретению; и (б) второй контейнер с содержащейся в нем композицией, причем указанная композиция содержит дополнительный цитотоксический или иной терапевтический агент. Готовое изделие в этом варианте осуществления может дополнительно содержать вкладыш в упаковку, на котором указано, что композиции можно применять для лечения конкретного патологического состояния. В альтернативном или дополнительном варианте готовое изделие может дополнительно содержать второй (или третий) контейнер, содержащий фармацевтически приемлемый буфер, такой как бактериостатическая вода для инъекций (БВДИ), фосфатно-солевой буферный раствор, раствор Рингера и раствор декстрозы. Оно также может содержать другие материалы, необходимые с коммерческой и пользовательской точки зрения, включая другие буферы, разбавители, фильтры, иглы и шприцы.
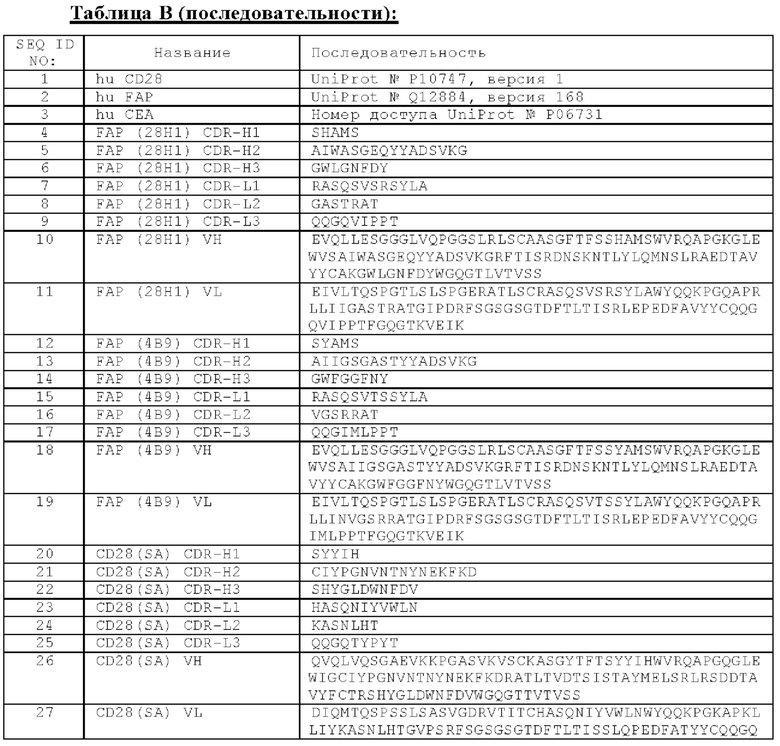
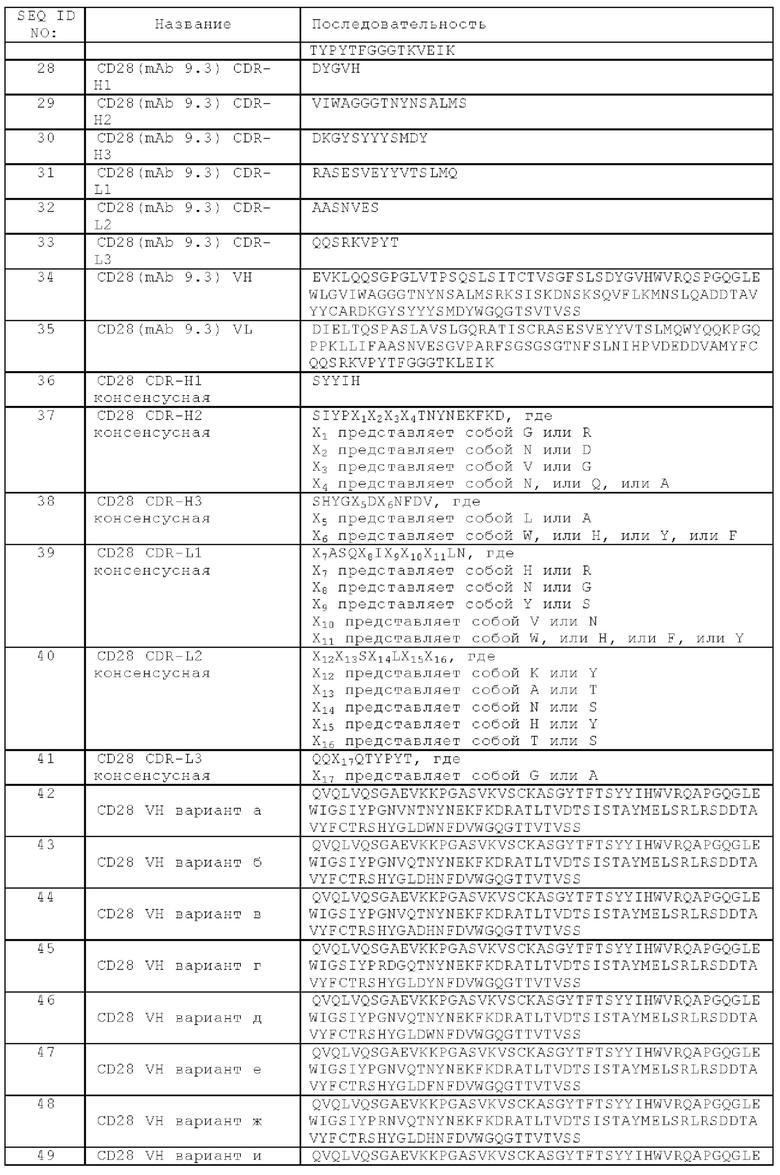
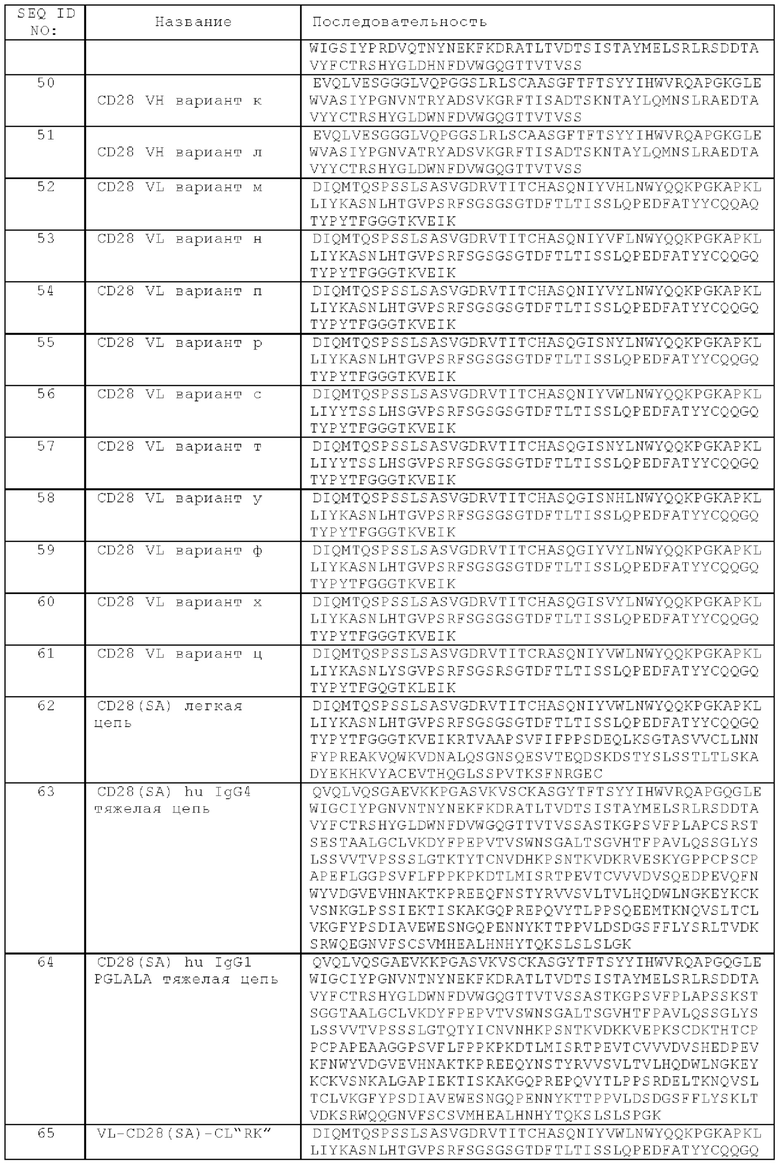

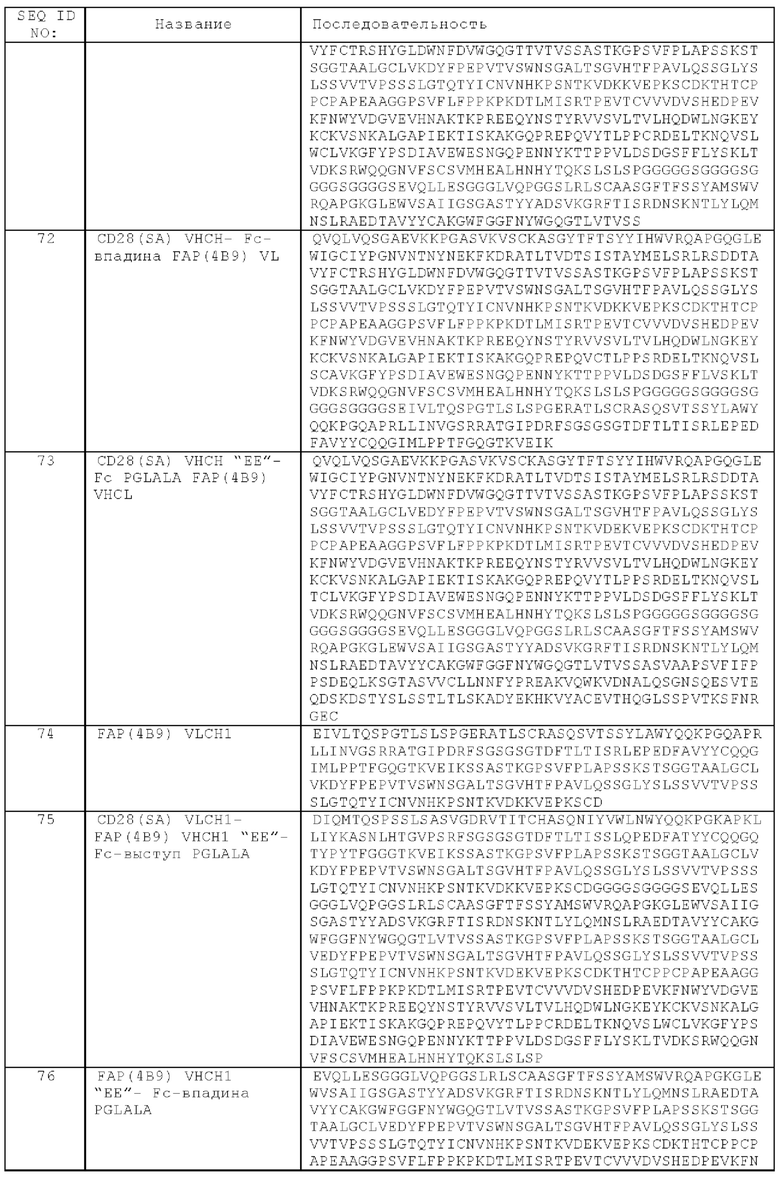
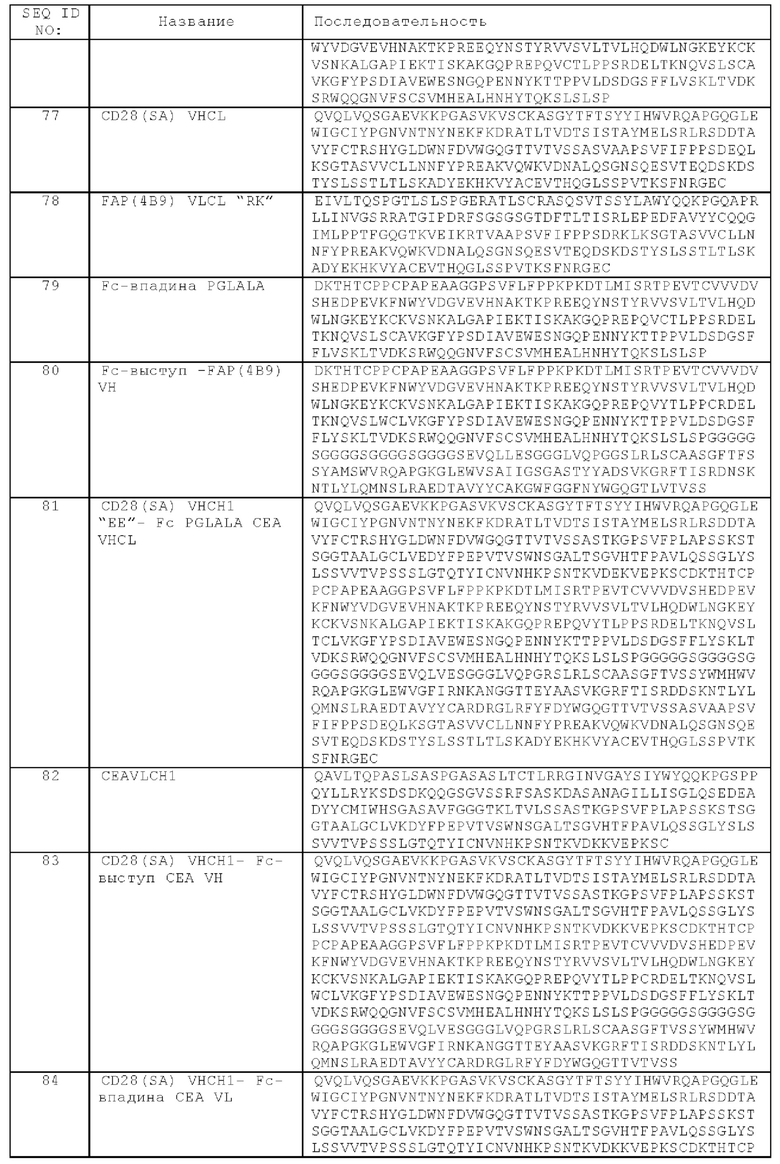



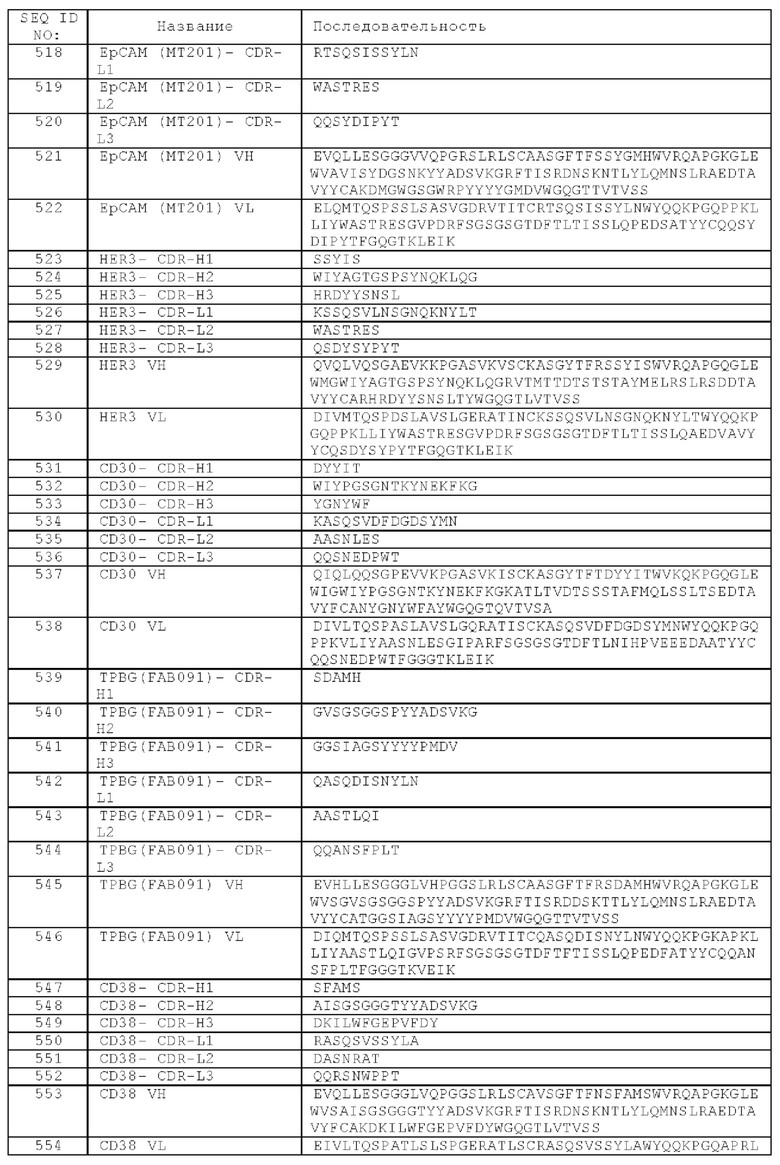
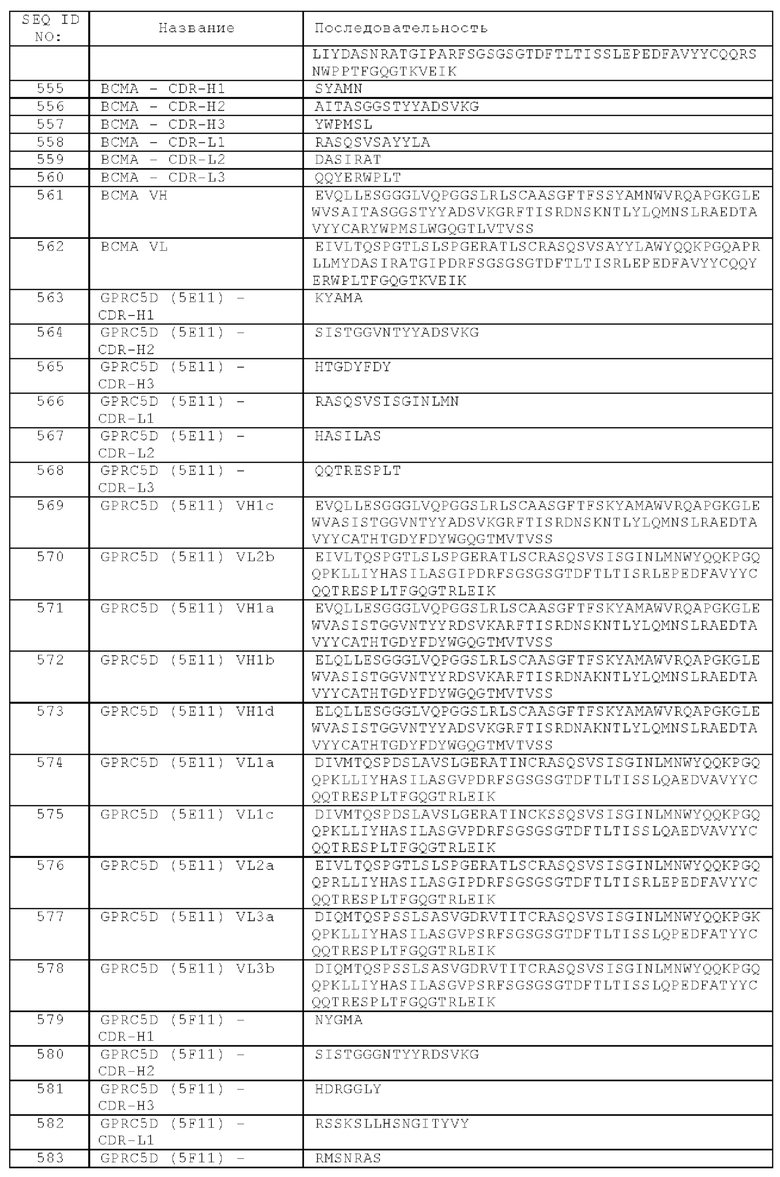
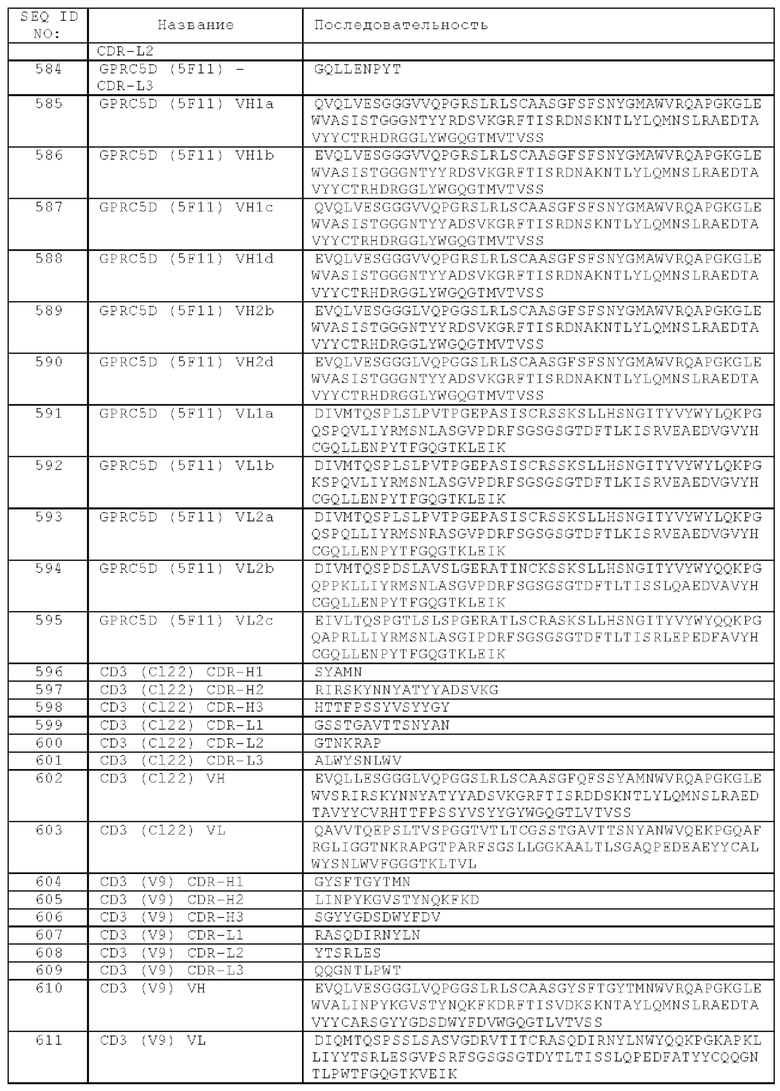
Общая информация в отношении нуклеотидных последовательностей легких и тяжелых цепей человеческих иммуноглобулинов приведена в: Kabat, Е.А., et al., Sequences of Proteins of Immunological Interest, 5th ed., Public Health Service, National Institutes of Health, Bethesda, MD (1991). Аминокислоты цепей антител пронумерованы и указаны в соответствии с системой нумерации в соответствии с Kabat (Kabat, Е.А., et al., Sequences of Proteins of Immunological Interest, 5th ed., Public Health Service, National Institutes of Health, Bethesda, MD (1991)), определенной выше.
Следующие пронумерованные параграфы (пар.) описывают аспекты настоящего изобретения:
1. Биспецифическая агонистическая CD28-антигенсвязывающая молекула, характеризующаяся одновалентным связыванием с CD28, содержащая
(а) один антигенсвязывающий домен, способный к специфическому связыванию с CD28,
(б) по меньшей мере один антигенсвязывающий домен, способный к специфическому связыванию с опухолеассоциированным антигеном, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
2. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по пар. 1, в которой Fc-домен принадлежит IgG, в частности представляет собой Fc-домен IgG1 или Fc-домен IgG4.
3. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по пар. 1 или 2, в которой Fc-домен принадлежит подклассу IgG1 человека и содержит аминокислотные мутации L234A, L235A и P329G (нумерация в соответствии с индексом EU по Kabat).
4. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пар. 1-3, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD28, содержит (i) вариабельную область тяжелой цепи (VHCD28), содержащую определяющую комплементарность область тяжелой цепи CDR-H1 с SEQ ID NO: 20, CDR-H2 с SEQ ID NO: 21 и CDR-H3 с SEQ ID NO: 22, и вариабельную область легкой цепи (VLCD28), содержащую определяющую комплементарность область легкой цепи CDR-L1 с SEQ ID NO: 23, CDR-L2 с SEQ ID NO: 24 и CDR-L3 с SEQ ID NO: 25; или
(ii) вариабельную область тяжелой цепи (VHCD28), содержащую CDR-H1 с SEQ ID NO: 36, CDR-H2 с SEQ ID NO: 37 и CDR-H3 с SEQ ID NO: 38, и вариабельную область легкой цепи (VLCD28), содержащую CDR-L1 с SEQ ID NO: 39, CDR-L2 с SEQ ID NO: 40 и CDR-L3 с SEQ ID NO: 41.
5. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пар. 1-4, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD28, содержит вариабельную область тяжелой цепи (VHCD28), содержащую CDR-H1 с SEQ ID NO: 20, CDR-H2 с SEQ ID NO: 21 и CDR-H3 с SEQ ID NO: 22, и вариабельную область легкой цепи (VLCD28), содержащую CDR-L1 с SEQ ID NO: 23, CDR-L2 с SEQ ID NO: 24 и CDR-L3 с SEQ ID NO: 25.
6. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пар. 15, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD28, содержит вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO:26, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO:27.
7. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пар. 1-4, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD28, содержит вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO:42, SEQ ID NO:43, SEQ ID NO:44, SEQ ID NO:45, SEQ ID NO:46, SEQ ID NO:47, SEQ ID NO:48, SEQ ID NO:49, SEQ ID NO:50 и SEQ ID NO:51, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO:27, SEQ ID NO:52, SEQ ID NO:53, SEQ ID NO:54, SEQ ID NO:55, SEQ ID NO:56, SEQ ID NO:57, SEQ ID NO:58, SEQ ID NO:59, SEQ ID N0:60 и SEQ ID N0:60.
8. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пар. 14 или 7, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD28, содержит
(а) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO:47, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO:54, или
(б) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO:47, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO:27, или
(в) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO:51, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO:61, или
(г) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO:46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO:53, или
(д) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO:46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO:54, или
(е) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO:46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO:59, или
(ж) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO:46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO:27, или
(и) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO:43, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO:27, или
(к) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO:42, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO:53, или
(л) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO:42, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO:59, или
(м) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO:42, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO:27.
9. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пар. 1-8, в которой антигенсвязывающий домен, способный к специфическому связыванию с опухолеассоциированным антигеном, представляет собой антигенсвязывающий домен, способный к специфическому связыванию с карциноэмбриональным антигеном (СЕА).
10. Биспецифическая CD28-антигенсвязывающая молекула по любому одному из пар. 1-9, причем антигенсвязывающий домен, способный к специфическому связыванию с СЕА, содержит вариабельную область тяжелой цепи (VHCEA), содержащую (i) CDR-H1, содержащую аминокислотную последовательность SEQ ID NO: 127, (ii) CDR-H2, содержащую аминокислотную последовательность SEQ ID NO: 128, и (iii) CDR-H3, содержащую аминокислотную последовательность SEQ ID NO: 129, и вариабельную область легкой цепи (VLCEA), содержащую (iv) CDR-L1, содержащую аминокислотную последовательность SEQ ID NO:130, (v) CDR-L2, содержащую аминокислотную последовательность SEQ ID NO: 131, и (vi) CDR-L3, содержащую аминокислотную последовательность SEQ ID NO: 132.
11. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пар. 1-10, в которой антигенсвязывающий домен, способный к специфическому связыванию с СЕА, содержит вариабельную область тяжелой цепи (VHCEA), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 133, и вариабельную область легкой цепи (VLCEA), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 134.
12. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пар. 1-8, в которой антигенсвязывающий домен, способный к специфическому связыванию с опухолеассоциированным антигеном, представляет собой антигенсвязывающий домен, способный к специфическому связыванию с белком активации фибробластов (FAP).
13. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пар. 1-8 или 12, в которой антигенсвязывающий домен, способный к специфическому связыванию с FAP, содержит
(а) вариабельную область тяжелой цепи (VHFAP), содержащую (i) CDR-H1, содержащую аминокислотную последовательность SEQ ID NO: 12, (ii) CDR-H2, содержащую аминокислотную последовательность SEQ ID NO: 13, и (iii) CDR-H3, содержащую аминокислотную последовательность SEQ ID NO: 14, и вариабельную область легкой цепи (VLFAP), содержащую (iv) CDR-L1, содержащую аминокислотную последовательность SEQ ID NO: 15, (v) CDR-L2, содержащую аминокислотную последовательность SEQ ID NO: 16, и (vi) CDR-L3, содержащую аминокислотную последовательность SEQ ID NO: 17, или (б) вариабельную область тяжелой цепи (VHFAP), содержащую (i) CDR-H1, содержащую аминокислотную последовательность SEQ ID NO:4, (ii) CDR-H2, содержащую аминокислотную последовательность SEQ ID NO:5, и (iii) CDR-H3, содержащую аминокислотную последовательность SEQ ID NO:6, и вариабельную область легкой цепи (VLFAP), содержащую (iv) CDR-L1, содержащую аминокислотную последовательность SEQ ID NO:7, (v) CDR-L2, содержащую аминокислотную последовательность SEQ ID NO:8, и (vi) CDR-L3, содержащую аминокислотную последовательность SEQ ID NO:9.
14. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пар. 1-8, или 12, или 13, в которой антигенсвязывающий домен, способный к специфическому связыванию с FAP, содержит (а) вариабельную область тяжелой цепи (VHFAP), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO:18, и вариабельную область легкой цепи (VLFAP), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO: 19, или
(б) вариабельную область тяжелой цепи (VHFAP), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO:10, и вариабельную область легкой цепи (VLFAP), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO:11.
15. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пар. 18, или 12, или 14, в которой антигенсвязывающий домен, способный к специфическому связыванию с FAP, содержит вариабельную область тяжелой цепи (VHFAP), содержащую аминокислотную последовательность SEQ ID NO:18, и вариабельную область легкой цепи (VLFAP), содержащую аминокислотную последовательность SEQ ID NO:19.
16. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пар. 1-15, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD28, представляет собой Fab-фрагмент или Kpocc-Fab-фрагмент.
17. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пар. 1-16, содержащая
(а) один Fab-фрагмент, способный к специфическому связыванию с CD28,
(б) один Kpocc-Fab-фрагмент, способный к специфическому связыванию с опухолеассоциированным антигеном, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
18. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пар. 1-16, содержащая
(а) первый Fab-фрагмент, способный к специфическому связыванию с CD28,
(б) второй Fab-фрагмент, способный к специфическому связыванию с опухолеассоциированным антигеном, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию,
причем первый Fab-фрагмент, способный к специфическому связыванию с CD28, слит на С-конце тяжелой цепи Fab с N-концом тяжелой цепи Fab второго Fab-фрагмента, способного к специфическому связыванию с опухолеассоциированным антигеном, который в свою очередь слит на своем С-конце с N-концом одной из субъединиц Fc-домена.
19. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пар. 1-16, содержащая
(а) первый Fab-фрагмент, способный к специфическому связыванию с CD28,
(б) второй и третий Fab-фрагменты, способные к специфическому связыванию с опухолеассоциированным антигеном, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию,
причем первый Fab-фрагмент, способный к специфическому связыванию с CD28, слит на С-конце тяжелой цепи Fab с N-концом тяжелой цепи Fab второго Fab-фрагмента, способного к специфическому связыванию с опухолеассоциированным антигеном, который в свою очередь слит на своем С-конце с N-концом первой субъединицы Fc-домена, и третий Fab-фрагмент, способный к специфическому связыванию с опухолеассоциированным антигеном, слит на С-конце тяжелой цепи Fab с N-концом второй субъединицы Fc-домена.
20. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пар. 1-16, содержащая
(а) Fab-фрагмент, способный к специфическому связыванию с CD28,
(б) домены VH и VL, способные к специфическому связыванию с опухолеассоциированным антигеном, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию,
причем Fab-фрагмент, способный к специфическому связыванию с CD28, слит на своем С-конце с N-концом первой субъединицы Fc-домена, и при этом один из доменов VH и VL, способных к специфическому связыванию с опухолеассоциированным антигеном, слит посредством пептидного линкера с С-концом первой субъединицы Fc-домена, а другой из доменов VH и VL, способных к специфическому связыванию с опухолеассоциированным антигеном, слит посредством пептидного линкера с С-концом второй субъединицы Fc-домена.
21. Полинуклеотид, кодирующий биспецифическую агонистическую CD28-антигенсвязывающую молекулу по любому из пар. 1-20.
22. Клетка-хозяин, содержащая полинуклеотид по пар. 21.
23. Способ получения биспецифической агонистической CD28-антигенсвязывающей молекулы по любому из пар. 1-20, включающий культивирование клетки-хозяина по пар. 22 в условиях, подходящих для экспрессии биспецифической антигенсвязывающей молекулы.
24. Фармацевтическая композиция, содержащая биспецифическую агонистическую CD28-антигенсвязывающую молекулу по любому из пар. 1-20 и по меньшей мере одно фармацевтически приемлемый эксципиент.
25. Фармацевтическая композиция по пар. 24 для применения в лечении рака.
26. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пар. 1-20 или фармацевтическая композиция по пар. 24 для применения в качестве лекарственного средства.
27. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пар. 1-20 или фармацевтическая композиция по пар. 24 для применения в лечении рака.
28. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пар. 1-20 для применения в лечении рака, причем агонистическую CD28-антигенсвязывающую молекулу вводят в комбинации с химиотерапевтическим агентом, лучевой терапией и/или другими агентами для применения в иммунотерапии рака.
29. Применение биспецифической агонистической CD28-антигенсвязывающей молекулы по любому из пар. 1-20 или фармацевтической композиции по пар. 24 в производстве лекарственного средства для лечения рака.
30. Способ ингибирования роста опухолевых клеток у субъекта, включающий введение субъекту эффективного количества биспецифической агонистической CD28-антигенсвязывающей молекулы по любому из пар. 1-20 или фармацевтической композиции по пар. 24 для ингибирования роста опухолевых клеток.
31. Способ лечения рака, включающий введение субъекту терапевтически эффективного количества биспецифической агонистической CD28-антигенсвязывающей молекулы по любому из пар. 1-20 или фармацевтической композиции по пар. 24.
32. Биспецифическая агонистическая CD28-антигенсвязывающая молекула, содержащая антигенсвязывающий домен, способный к специфическому связыванию с CD28, антигенсвязывающий домен, способный к специфическому связыванию с поверхностным антигеном В-клеток, и Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
33. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по пар. 32, характеризующаяся одновалентным связыванием с CD28.
34. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по пар. 32, дополнительно характеризующаяся одновалентным связыванием с поверхностным антигеном В-клеток.
35. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пар. 32-34, в которой Fc-домен принадлежит подклассу IgG1 человека и содержит аминокислотные мутации L234A, L235A и P329G (нумерация в соответствии с индексом EU по Kabat).
36. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пар. 32-35, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD28, содержит
(i) вариабельную область тяжелой цепи (VHCD28), содержащую определяющую комплементарность область тяжелой цепи CDR-H1 с SEQ ID NO:20, CDR-H2 с SEQ ID NO:21 и CDR-H3 с SEQ ID NO:22, и вариабельную область легкой цепи (VLCD28), содержащую определяющую комплементарность область легкой цепи CDR-L1 с SEQ ID NO:23, CDR-L2 с SEQ ID NO:24 и CDR-L3 с SEQ ID NO:25; или
(ii) вариабельную область тяжелой цепи (VHCD28), содержащую CDR-H1 с SEQ ID NO:36, CDR-H2 с SEQ ID NO:37 и CDR-H3 с SEQ ID NO:38, и вариабельную область легкой цепи (VLCD28), содержащую CDR-L1 с SEQ ID NO:39, CDR-L2 с SEQ ID NO:40 и CDR-L3 с SEQ ID NO:41.
37. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пар. 32-36, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD28, содержит вариабельную область тяжелой цепи (VHCD28), содержащую CDR-H1 с SEQ ID NO:20, CDR-H2 с SEQ ID NO:21 и CDR-H3 с SEQ ID NO:22, и вариабельную область легкой цепи (VLCD28), содержащую CDR-L1 с SEQ ID NO:23, CDR-L2 с SEQ ID NO:24 и CDR-L3 с SEQ ID NO:25.
38. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пар. 32-37, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD28, содержит вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO:26, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 96%, 97%, 98%, 99% или 100% идентичной аминокислотной последовательности SEQ ID NO:27.
39. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пар. 32-36, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD28, содержит вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO:42, SEQ ID NO:43, SEQ ID NO:44, SEQ ID NO:45, SEQ ID NO:46, SEQ ID NO:47, SEQ ID NO:48, SEQ ID NO:49, SEQ ID N0:50 и SEQ ID NO:51, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO:27, SEQ ID NO:52, SEQ ID NO:53, SEQ ID NO:54, SEQ ID NO:55, SEQ ID NO:56, SEQ ID NO:57, SEQ ID NO:58, SEQ ID NO:59, SEQ ID NO:60 и SEQ ID NO:61.
40. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пар. 32-36 или 39, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD28, содержит
(а) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO:47, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO:54, или
(б) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO:47, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO:27, или
(в) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO:51, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO:61, или
(г) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO:48, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO:53, или
(д) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO:48, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO:54, или
(е) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO:48, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO:59, или
(ж) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO:48, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO:27, или
(и) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO:43, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO:27, или
(к) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO:42, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO:53, или
(л) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO:42, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO:59, или
(м) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO:42, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO:27.
41. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пар. 32-36, или 39, или 40, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD28, содержит вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO:46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO:53, или вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO:47, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO:54, или вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO:47, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO:9, или вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 16, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO:9.
42. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пар. 32-41, в которой поверхностный антиген В-клеток выбран из группы, состоящей из CD19, CD79b, CD20, CD22 и CD37.
43. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пар. 32 42, в которой антигенсвязывающий домен, способный к специфическому связыванию с поверхностным антигеном В-клеток, представляет собой антигенсвязывающий домен, способный к специфическому связыванию с CD19.
44. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пар. 32-43, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD 19, содержит
(а) вариабельную область тяжелой цепи (VHCD19), содержащую (i) CDR-H1, содержащую аминокислотную последовательность SEQ ID NO:406, (ii) CDR-H2, содержащую аминокислотную последовательность SEQ ID NO:407, и (iii) CDR-Н3, содержащую аминокислотную последовательность SEQ ID NO:408, и вариабельную область легкой цепи (VLCD19), содержащую (iv) CDR-L1, содержащую аминокислотную последовательность SEQ ID NO:409, (v) CDR-L2, содержащую аминокислотную последовательность SEQ ID NO:410, и (vi) CDR-L3, содержащую аминокислотную последовательность SEQ ID NO:411, или
(б) вариабельную область тяжелой цепи (VHCD19), содержащую (i) CDR-H1, содержащую аминокислотную последовательность SEQ ID NO:414, (ii) CDR-H2, содержащую аминокислотную последовательность SEQ ID NO:415, и (iii) CDR-Н3, содержащую аминокислотную последовательность SEQ ID NO:416, и вариабельную область легкой цепи (VLCD19), содержащую (iv) CDR-L1, содержащую аминокислотную последовательность SEQ ID NO:417, (v) CDR-L2, содержащую аминокислотную последовательность SEQ ID NO:418, и (vi) CDR-L3, содержащую аминокислотную последовательность SEQ ID NO:419.
45. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пар. 32-44, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD 19, содержит (а) вариабельную область тяжелой цепи (VHCD19), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 98% или 100% идентичной аминокислотной последовательности SEQ ID NO:412, и вариабельную область легкой цепи (VLCD19), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 98% или 100% идентичной аминокислотной последовательности SEQ ID NO:413, или
(б) вариабельную область тяжелой цепи (VHCD19), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 98% или 100% идентичной аминокислотной последовательности SEQ ID NO:420, и вариабельную область легкой цепи (VLCD19), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 98% или 100% идентичной аминокислотной последовательности SEQ ID NO:421.
46. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пар. 32 45, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD19, содержит вариабельную область тяжелой цепи (VHCD19), содержащую аминокислотную последовательность SEQ ID NO:412, и вариабельную область легкой цепи (VLCD19), содержащую аминокислотную последовательность SEQ ID NO:413.
47. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пар. 32-43, в которой антигенсвязывающий домен, способный к специфическому связыванию с поверхностным антигеном В-клеток, представляет собой антигенсвязывающий домен, способный к специфическому связыванию с CD79b.
48. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пар. 32-43 или 47, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD79b, содержит вариабельную область тяжелой цепи (VHCD79b), содержащую (i) CDR-H1, содержащую аминокислотную последовательность SEQ ID NO:422, (ii) CDR-H2, содержащую аминокислотную последовательность SEQ ID NO:423, и (iii) CDR-Н3, содержащую аминокислотную последовательность SEQ ID NO:424, и вариабельную область легкой цепи (VLCD79b), содержащую (iv) CDR-L1, содержащую аминокислотную последовательность SEQ ID NO:425, (v) CDR-L2, содержащую аминокислотную последовательность SEQ ID NO:426, и (vi) CDR-L3, содержащую аминокислотную последовательность SEQ ID NO:427.
49. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пар. 32-43, или 47, или 48, в которой антигенсвязывающий домен, способный к специфическому связыванию с CD79b, содержит вариабельную область тяжелой цепи (VHCD79b), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 98% или 100% идентичной аминокислотной последовательности SEQ ID NO:428, и вариабельную область легкой цепи (VLCD79b), содержащую аминокислотную последовательность, которая является по меньшей мере на около 95%, 98% или 100% идентичной аминокислотной последовательности SEQ ID NO:429.
50. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пар. 32-49, содержащая
(а) один Fab-фрагмент, способный к специфическому связыванию с CD28,
(б) один Kpocc-Fab-фрагмент, способный к специфическому связыванию с поверхностным антигеном В-клеток, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
51. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пар. 32-49, содержащая
(а) первый Fab-фрагмент, способный к специфическому связыванию с CD28,
(б) второй Fab-фрагмент, способный к специфическому связыванию с поверхностным антигеном В-клеток, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию,
причем первый Fab-фрагмент, способный к специфическому связыванию с CD28, слит на С-конце тяжелой цепи Fab с N-концом тяжелой цепи Fab второго Fab-фрагмента, способного к специфическому связыванию с опухолеассоциированным антигеном, который в свою очередь слит на своем С-конце с N-концом одной из субъединиц Fc-домена.
52. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пар. 32-49, содержащая
(а) первый Fab-фрагмент, способный к специфическому связыванию с CD28,
(б) второй и третий Fab-фрагменты, способные к специфическому связыванию с поверхностным антигеном В-клеток, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию,
причем первый Fab-фрагмент, способный к специфическому связыванию с CD28, слит на С-конце тяжелой цепи Fab с N-концом тяжелой цепи Fab второго Fab-фрагмента, способного к специфическому связыванию с опухолеассоциированным антигеном, который в свою очередь слит на своем С-конце с N-концом первой субъединицы Fc-домена, и третий Fab-фрагмент, способный к специфическому связыванию с опухолеассоциированным антигеном, слит на С-конце тяжелой цепи Fab с N-концом второй субъединицы Fc-домена.
53. Фармацевтическая композиция, содержащая биспецифическую агонистическую CD28-антигенсвязывающую молекулу по любому из пар. 32-52 и по меньшей мере одно фармацевтически приемлемый эксципиент.
54. Полинуклеотид, кодирующий биспецифическую агонистическую CD28-антигенсвязывающую молекулу по любому из пар. 32-52.
55. Вектор, содержащий полинуклеотид по пар. 54.
56. Клетка-хозяин, содержащая вектор по пар. 55 или полинуклеотид по пар. 54.
57. Способ получения биспецифической агонистической CD28-антигенсвязывающей молекулы по любому из пар. 32-52, включающий культивирование клетки-хозяина по пар. 25 в условиях, подходящих для экспрессии биспецифической антигенсвязывающей молекулы.
58. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пар. 32-52 или фармацевтическая композиция по пар. 53 для применения в качестве лекарственного средства.
59. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пар. 32-52 или фармацевтическая композиция по пар. 53 для применения в лечении рака.
60. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пар. 32-52 для применения в лечении рака, причем агонистическая CD28-антигенсвязывающая молекула предназначена для применения в комбинации с химиотерапевтическим агентом, лучевой терапией и/или другими агентами для применения в иммунотерапии рака.
60. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пар. 32-52 для применения в лечении рака, причем агонистическая CD28-антигенсвязывающая молекула предназначена для применения в комбинации с активирующим Т-клетки биспецифическим антителом к CD3.
61. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пар. 32-52 для применения в лечении рака, причем агонистическая CD28-антигенсвязывающая молекула предназначена для применения в комбинации с анти-CD20/анти-CD3 биспецифическим антителом.
62. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пар. 32-52 для применения в лечении рака, причем агонистическая CD28-антигенсвязывающая молекула предназначена для применения в комбинации с агентом, блокирующим взаимодействие PD-L1/PD-1.
63. Применение биспецифической агонистической CD28-антигенсвязывающей молекулы по любому из пар. 32-52 или фармацевтической композиции по пар. 53 в производстве лекарственного средства для лечения рака.
64. Способ ингибирования роста опухолевых клеток у субъекта, включающий введение субъекту эффективного количества биспецифической агонистической CD28-антигенсвязывающей молекулы по любому из пар. 32-52 или фармацевтической композиции по пар. 53 для ингибирования роста опухолевых клеток.
65. Способ лечения рака, включающий введение субъекту терапевтически эффективного количества биспецифической агонистической CD28-антигенсвязывающей молекулы по любому из пар. 32-52 или фармацевтической композиции по пар. 53.
ПРИМЕРЫ
Ниже приведены примеры способов и композиций по изобретению. Следует понимать, что на практике можно реализовать ряд других вариантов осуществления с учетом общего описания, приведенного выше.
Технологии рекомбинантных ДНК
Для манипуляций с ДНК применяли стандартные методы, описанные в Sambrook et al., Molecular cloning: A laboratory manual; Cold Spring Harbor Laboratory Press, Cold Spring Harbor, New York, 1989. Молекулярные биологические реагенты использовали в соответствии с инструкциями производителя. Общая информация в отношении нуклеотидных последовательностей легких и тяжелых цепей человеческого иммуноглобулина приведена в: Kabat, Е.А. et al., (1991) Sequences of Proteins of Immunological Interest, Fifth Ed., NIH Publication No 91-3242.
Секвенирование ДНК
Последовательности ДНК определяли с помощью секвенирования двух цепей.
Синтез генов
Необходимые генные сегменты, при необходимости, были созданы с помощью ПЦР с применением соответствующих матриц или были синтезированы Geneart AG (Regensburg, Germany) или Genscript (New Jersey, USA) из синтетических олигонуклеотидов и продуктов ПЦР с помощью автоматизированного синтеза генов. Генные сегменты, фланкируемые сингулярными сайтами расщепления рестрикционной эндонуклеазой, клонировали в стандартные векторы для клонирования/секвенирования. Плазмидную ДНК очищали из трансформированных бактерий, а концентрацию определяли с помощью УФ-спектроскопии. Последовательность ДНК субклонированных генных фрагментов подтверждали с помощью секвенирования ДНК. Генные сегменты конструировали с подходящими рестрикционными сайтами, чтобы обеспечить возможность субклонирования в соответствующие экспрессионные векторы. Все конструкции конструировали с 5'-концевой последовательностью ДНК, кодирующей лидерный пептид, который направляет белки для секреции в эукариотических клетках.
Методики клеточного культивирования
Использовали стандартные методики клеточного культивирования, как описано в Current Protocols in Cell Biology (2000), Bonifacino, J.S., Dasso, M., Harford, J.В., Lippincott-Schwartz, J. and Yamada, K.M. (eds.), John Wiley & Sons, Inc.
Очистка белка
Белки очищали из профильтрованных клеточных культуральных супернатантов согласно стандартным протоколам. Вкратце, антитела наносили на сефарозную колонку с протеином A (GE healthcare) и промывали ФСБ. Элюирование антител проводили при рН 2,8 с последующей незамедлительной нейтрализацией образца. Агрегированный белок отделяли от мономерных антител с помощью эксклюзионной хроматографии (Superdex 200, GE Healthcare) в ФСБ или в 20 мМ гистидине, 150 мМ NaCl, рН 6,0. Фракции мономерных антител объединяли, концентрировали (при необходимости), используя, например, центрифужный концентратор MILLIPORE Amicon Ultra (30 НОММ), замораживали и хранили при -20°С или -80°С. Часть образцов предоставляли для последующего белкового анализа и аналитического определения, например, с помощью ДСН-ПААГ, эксклюзионной хроматографии (ЭХ) или масс-спектрометрии.
ДСН-ПААГ
Гель-систему NuPAGE® Pre-Cast (Invitrogen) использовали в соответствии с инструкцией производителя. В частности, использовали 10% или 4-12% гели NuPAGE® Novex® Bis-TRIS Pre-Cast (рН 6,4) и NuPAGE® MES (восстановленные гели с добавление антиоксидантного подвижного буфера NuPAGE®) или подвижный буфер MOPS (невосстановленные гели).
Аналитическая эксклюзионная хроматография
Эксклюзионную хроматографию (ЭХ) для определения агрегации и олигомерного состояния антител проводили с помощью ВЭЖХ-хроматографии. Вкратце, очищенные с протеином А антитела наносили на колонку Tosoh TSKgel G3000SW в 300 мМ NaCl, 50 мМ KH2PO4/K2HPO4, рН 7,5, на системе Agilent НРГС 1100 или на колонку Superdex 200 (GE Healthcare) в 2 х ФСБ на ВЭЖХ-системе Dionex. Количественную оценку элюированного белка проводили по УФ-поглощению и интеграции площадей пиков. Стандартом служил стандарт гель-фильтрации BioRad 151 1901.
Масс-спектрометрия
В этом разделе описано определение характеристик мультиспецифических антител с обменом VH/VE (CrossMab VH/VE) с акцентом на их правильную сборку. Ожидаемые первичные структуры анализировали с помощью масс-спектрометрии с ионизацией электрораспылением (ИЭР-МС) дегликозилированных интактных CrossMab и дегликозилированных/расщепленных плазмином или альтернативным образом дегликозилированных/расщепленных ограниченным LysC CrossMab.
VH/VL CrossMab дегликозилировали N-гликозидазой F в фосфатном или Трис-буфере при 37°С в течение до 17 ч при концентрации белка 1 мг/мл. расщепление плазмином или ограниченным LysC (Roche) проводили с 100 мкг дегликозилированных VH/VL CrossMab в Трис-буфере при рН 8 при комнатной температуре в течение 120 часов и при 37°С в течение 40 мин, соответственно. Перед масс-спектрометрией образцы обессоливали посредством ВЭЖХ на колонке Sephadex G25 (GE Healthcare). Общую массу определяли с помощью ИЭР-МС на системе maXis 4G UHR-QTOF MS (Bruker Daltonik), оснащенной источником TriVersa NanoMate (Advion).
Определение связывания и аффинности связывания мультиспецифических антител с соответствующими антигенами с помощью поверхностного плазмонного резонанса (ППР) (BIACORE)
Связывание созданных антител с соответствующими антигенами исследуют методом поверхностного плазмонного резонанса, используя инструмент BIACORE (GE Healthcare Biosciences AB, Uppsala, Sweden). Вкратце, для измерений аффинности козьи античеловеческие антитела IgG, JIR 109-005-098 иммобилизуют на чипе СМ5 посредством сопряжения аминов для представления антител против соответствующего антигена. Связывание измеряют в буфере HBS (HBS-P (10 мМ ГЭПЭС, 150 мМ NaCl, 0,005% Твин 20, ph 7,4), 25°С (или, в альтернативном варианте, 37°С). Антиген (R&D Systems или лабораторный, очищенный) добавляли в раствор в различных концентрациях. Ассоциацию измеряли путем введения антигена в течение от 80 секунд до 3 минут; диссоциацию измеряли путем промывания поверхности чипа буфером HBS в течение 3-10 минут, а значение KD оценивали, используя 1:1 модель связывания Лэнгмюра. Данные отрицательного контроля (например, кривые для буфера) вычитают из кривых для образцов для коррекции на характерный системный дрейф базовой линии и для снижения сигнала шума. Для анализа сенсограмм и для расчета данных аффинности используют соответствующее оценочное программное обеспечение Biacore.
Пример 1
Создание и выработка биспецифических антигенсвязывающих молекул, нацеленных на CD28 и белок активации фибробластов (FAP) или карциноэмбриональный антиген (СЕА)
1.1 Клонирование биспецифических антигенсвязывающих молекул, нацеленных на CD28 и белок активации фибробластов (FAP) или карциноэмбриональный антиген (СЕА)
Клонирование антигена:
Фрагмент ДНК, кодирующий внеклеточный домен (аминокислоты от 1 до 134 зрелого белка) CD28 человека (Uniprot: Р10747) вставляли в рамке в два разных реципиентных вектора млекопитающих выше фрагмента, кодирующего гуманизированным Fc-фрагмент IgG1, который служит тегом растворимости и очистки. Один из экспрессионных векторов содержал в Fc-области мутации типа «впадина», другой - мутации типа «выступ», а также С-концевой avi-тег (GLNDIFEAQKIEWHE, SEQ ID NO:387), обеспечивающий возможность специфического биотинилирования во время коэкспрессии с биотинлигазой Bir А. Кроме того, оба Fc-фрагмента содержали мутации PG-LALA. Оба вектора совместно трансфицировали в комбинации с плазмидой, кодирующей биотинлигазу BirA, чтобы получить димерную конструкцию CD28-Fc с одновалентным биотинилированным avi-тегом С-конце цепи Fc-выступа.
Вариабельные домены клона FAP 4 В9, связывающую СЕА молекулу и клоны CD28 SA и mAb 9.3 использовали для создания различных нацеленных на опухоль конструкций CD28. Создание и подготовка клона FAP 4 В9 описаны в WO 2012/020006 А2, которая включена в данный документ посредством ссылки. Клон СЕА, используемый в молекулах, описан в WO 2007/071422, а суперагонистическое (SA) антитело CD28 с VH, содержащей аминокислотную последовательность SEQ ID NO:26, и VL, содержащей аминокислотную последовательность SEQ ID NO:27, описано в WO 2006/050949. Описание антитела mAb 9.3 можно найти в Tan et al. J. Immunology 2002, 169, 1119-1125. Для создания соответствующих экспрессионных плазмид использовали последовательности соответствующих вариабельных доменов, которые субклонировали в рамке с соответствующими константными областями, которые были предварительно вставлены в соответствующий реципиентный экспрессионный вектор млекопитающих. Схематическое описание полученных в результате молекул приведено на Фиг. 1А-1П. Где указано, мутации Pro329Gly, Leu234A1a и Leu235Ala (PG-LALA) были внесены в константную область тяжелых цепей IgG1 человека для устранения связывания с Fc-гамма-рецепторами. Для создания несимметричных биспецифических антител Fc-фрагменты содержали мутации типа «выступ» или «впадина», чтобы предотвратить неправильное спаривание тяжелых цепей. Чтобы предотвратить неправильное спаривание легких цепей в би- и мультиспецифических конструкциях антител, в один связывающий фрагмент вносили обмен доменов VH/VL или СН1/С-каппа (технология CrossFab). В другом связывающем фрагменте в домены СН1 и С-каппа вносили заряды.
Клонировали следующие молекулы, схематические иллюстрации которых показаны на Фиг. 1А-1П:
Молекула A: CD28(SA) (hu IgG4), TGN1412, CD28 (SA) антитело в изотипе IgG4 человека (Фиг. 1А), содержит аминокислотные последовательности SEQ ID NO:62 и SEQ ID NO:63 (P1AE1975).
Молекула В: CD28(SA) (PG-EALA), CD28 (SA) антитело в изотипе huIgG1 PG-LALA (Фиг. 1Б) содержит аминокислотные последовательности SEQ ID NO:62 и SEQ ID NO:64 (P1AD9289).
Молекула С: FAP(4B9)-CD28(SA) формат 1+1, биспецифическая huIgGI PG-LALA молекула кросс-Fab с заряженными модификациями в CD28(SA) Fab-фрагменте (выступ) и обменом VH/VL в FAP(4B9) Fab-фрагменте (впадина) (Фиг. 1 В), содержащая аминокислотные последовательности SEQ ID NO:65, 66, 67 и 68 (P1AD4492).
Молекула D: FAP(4B9)-CD28(SA) формат 1+4, биспецифическая четырехвалентная анти-CD28 (SA) и одновалентная анти-FAP huIgGI PG-LALA конструкция. Домены VH и VL клона FAP 4 В9 сливали с С-концом соответствующих цепей Fc-домена (VH: цепь выступа, VL: цепь впадины) (Фиг. 1Е). Молекула содержит аминокислотные последовательности из SEQ ID NO: 62, 69 и 70 (P1AD9018).
Молекула Е: FAP(4B9)-CD28(SA) формат 1+2, биспецифическая двухвалентная анти-CD28 (SA) и одновалентная анти-FAP huIgGI PG-LALA конструкция. Домены VH и VL клона FAP 4 В9 сливали с С-концом соответствующих цепей Fc-домена (VH: цепь выступа, VL: цепь впадины) (Фиг. 1Г). Молекула содержит аминокислотные последовательности из SEQ ID NO:62, 71 и 72 (P1AD9011).
Молекула F: FAP(4B9)-CD28(SA) 2+2, биспецифическая двухвалентная анти-CD28 (SA) и двухвалентная анти-FAP huIgG1 PG-LALA конструкция кросс-Fab, заряженные модификации в анти-CD28 Fab-фрагментах, слияние VH анти-FAP Kpocc-Fab-фрагментов с обменом СН1/С-каппа с С-концом Fc-фрагмента (Фиг. 1Д). Молекула содержит аминокислотные последовательности из SEQ ID NO:65, 73 и 74 (P1AD4493).
Молекула G: FAP (4B9)-CD28 (SA) 2+1, биспецифическая одновалентная анти-CD28 (SA) и двухвалентная анти-FAP huIgG1 PG-LALA конструкция кросс-Fab, «классическая ориентация», обмен VH/VL в анти-CD28 кросс-Fab-фрагменте, заряженная модификация в анти-FAP Fab-фрагментах (Фиг. 1Н). Молекула содержит аминокислотные последовательности из SEQ ID NO:75, 76, 77 и 78 (P1AD5231).
Молекула Н: FAP(4B9) - CD28(SA) С-01, 1+1 биспецифическая одновалентная анти-CD28 (SA) и одновалентная анти-FAP huIgG1 PG-LALA молекула кросс-Fab, «голова к хвосту», обмен VH/VL в анти-CD28 кросс-Fab-фрагменте, заряженная модификация в анти-FAP связывающей молекуле (Фиг. 1П). Молекула содержит аминокислотные последовательности из SEQ ID NO:75, 77, 78 и 79 (Р1АЕ2021).
Молекула I: FAP(4B9) - CD28(SA) С-04, 1+1 биспецифическая одновалентная анти-CD28 (SA) и одновалентная анти-FAP huIgG1 PG-LALA конструкция. Домены VH и VL связывающей FAP молекулы 4 В9 сливали с С-концом соответствующих цепей Fc-фрагмента (VH: цепь выступа, VL: цепь впадины) (Фиг. 1М). Молекула содержит аминокислотные последовательности из SEQ ID NO:62, 72 и 80 (Р1АЕ2236).
Молекула J: CEA-CD28(SA) 2+2, биспецифическая двухвалентная анти-CD28 (SA) и двухвалентная анти-СЕА huIgG1 PG-LALA конструкция кросс-Fab, заряженные модификации в анти-CD28 Fab-фрагментах, слияние VH анти-СЕА Kpocc-Fab-фрагмента с обменом СН1/С-каппа с С-концом Fc-фрагмента (Фиг. 1И). Молекула содержит аминокислотные последовательности из SEQ ID NO:65, 81 и 82 (Р1АЕ1195).
Молекула К: CEA-CD28(SA) 1+2, биспецифическая двухвалентная анти-CD28 (SA) и одновалентная анти-СЕА huIgG1 PG-LALA конструкция. Домены VH и VL связывающей СЕА молекулы сливали с С-концом соответствующих цепей Fc-фрагмента (VH: цепь выступа, VL: цепь впадины) (Фиг. 1Ж). Молекула содержит аминокислотные последовательности из SEQ ID NO:62, 83 и 84 (Р1АЕ1194).
Молекула L: одновалентная IgG CD28 (SA), одновалентная анти-CD28 (SA) hulgG1 PG-LALA конструкция, в которой тяжелая цепь CD28 экспрессируется в виде Fc-цепи «впадины» в комбинации с Fc-фрагментом (выступ) (Фиг. 1К). Молекула содержит аминокислотные последовательности из SEQ ID NO:65, 85 и 86 (P1AD8944).
Молекула М: CEA-CD28(SA) формат 1+1, биспецифическая hulgGI PG-LALA молекула кросс-Fab с заряженными модификациями в CD28(SA) Fab-фрагменте (выступ) и обменом VH/VL в СЕА Kpocc-Fab-фрагменте (впадина) (Фиг. 1Л), содержащая аминокислотные последовательности SEQ ID NO:65, 66, 87 и 88 (Р1АЕ3127).
Молекула N: mab 9.3 (PG-LALA), клон mAb9.3 в изотипе IgG1 PG-LALA человека (как на Фиг. 1Б). Молекула содержит аминокислотные последовательности из SEQ ID NO:89 и 90 (P1AD5142).
Молекула О: FAP(4B9) - CD28(mAb9.3) С-03, биспецифическая huIgG1 PG-LALA кросс-Fab конструкция с заряженными модификациями в mAb9.3 Fab-фрагменте (выступ) и обменом VH/VL в анти-FAP фрагмента (впадина) (как на Фиг. 1 В). Молекула содержит аминокислотные последовательности из SEQ ID NO:67, 68, 91 и 92 (Р1АЕ2238).
Молекула Р: FAP(4B9)-CD28(mAb9.3) 1+4, биспецифическая четырехвалентная анти-CD28 mAb9.3 и анти-FAP huIgG14 PG-LALA конструкция. Домены VH и VL связывающей FAP молекулы сливали с С-концом соответствующих цепей Fc-фрагмента (VH: цепь выступа, VL: цепь впадины) (как на Фиг. 1Е). Молекула содержит аминокислотные последовательности из SEQ ID NO:89, 93 и 94 (P1AD8969).
Молекула Q: FAP(4B9)-CD28(mAb9.3) 1+2, биспецифическая двухвалентная анти-CD28 тАЬ9.3 и одновалентная анти-FAP huIgG1 PG-LALA конструкция. Домены VH и VL связывающей FAP молекулы сливали с С-концом соответствующих цепей Fc-фрагмента (VH: цепь выступа, VL: цепь впадины) (как на Фиг. 1Г). Молекула содержит аминокислотные последовательности из SEQ ID NO:89, 95 и 96 (P1AD8962).
Молекула R: FAP(4B9)-CD28(mAb9.3) 2+2, биспецифическая двухвалентная анти-CD28 mAb9.3 и двухвалентная анти-FAP huIgG1 PG-LALA конструкция кросс-Fab, заряженные модификации в mAb9.3 FAP фрагменте, слияние VH анти-FAP Fab-фрагмента с обменом СН1/С-каппа кросс-Fab с С-концом Fc-фрагмента (как на Фиг. 1Д). Молекула содержит аминокислотные последовательности из SEQ ID NO:97, 98 и 99 (P1AD8968).
Молекула S: FAP (4B9)-CD28(mAb9.3) 2+1, биспецифическая одновалентная анти-CD28 (mAb9.3) и двухвалентная анти-FAP huIgG1 PG-LALA конструкция кросс-Fab, «классическая ориентация», обмен VH/VL в анти-CD28 (mAb9.3) Kpocc-Fab-фрагменте, заряженная модификация в анти-FAP Fab-фрагментах (как на Фиг. 1Н). Молекула содержит аминокислотные последовательности из SEQ ID NO:76, 77, 100 и 101 (P1AD5560).
Молекула Т: FAP(4B9) - CD28(mAb9.3) С-02, биспецифическая одновалентная анти-CD28 (тАЬ9.3) и одновалентная анти-FAP huIgG1 PG-LALA конструкция кросс-Fab, «голова к хвосту», обмен VH/VL в анти-CD28 (mAb9.3) Kpocc-Fab-фрагменте, заряженная модификация в анти-FAP фрагменте (как на Фиг. 1П). Молекула содержит аминокислотные последовательности из SEQ ID NO:78, 79, 100 и 101 (Р1АЕ2022).
Молекула U: FAP(4B9) - CD28(mAb9.3) С-05, биспецифическая одновалентная анти-CD28 (mAb9.3) и одновалентная анти-FAP hulgGI PG-LALA конструкция. Домены VH и VL связывающей FAP молекулы 4 В9 сливали с С-концом соответствующих цепей Fc-фрагмента (VH: цепь выступа, VL: цепь впадины) (как на Фиг. 1М). Молекула содержит аминокислотные последовательности из SEQ ID NO:80, 89 и 96 (Р1АЕ2237).
Молекула V: CEA-CD28(mAb9.3) 2+2, биспецифическая двухвалентная анти-CD28 (mAb9.3) и двухвалентная анти-СЕА huIgG1 PG-LALA конструкция кросс-Fab, заряженные модификации в mAb9.3 Fab-фрагменте, слияние VH анти-СЕА Kpocc-Fab-фрагмента с обменом СН1/С-каппа с С-концом Fc-фрагмента (как на Фиг. 1И). Молекула содержит аминокислотные последовательности из SEQ ID NO:82, 89 и 102 (Р1АЕ1193).
Молекула W: CEA-CD28(mAb9.3) 1+2, биспецифическая двухвалентная анти-CD28 (mAb9.3) и одновалентная анти-СЕА huIgG1 PG-LALA конструкция. Домены VH и VL связывающей СЕА молекулы сливали с С-концом соответствующих цепей Fc-фрагмента (VH: цепь выступа, VL: цепь впадины) (как на Фиг. 1Ж). Молекула содержит аминокислотные последовательности из SEQ ID NO:89, 103 и 104 (Р1АЕ1192).
Молекула X: одновалентный IgG CD28 (mAb9.3), в котором тяжелая цепь CD28 экспрессируется в виде Fc-цепи «впадины» в комбинации с Fc-фрагментом (выступ) (как на Фиг. 1К). Молекула содержит аминокислотные последовательности из SEQ ID NO:86, 105 и 106 (P1AD8938).
Кроме того, готовили триспецифическую молекулу:
Молекула Y, FAP (4B9)-CD28(TGN1412)-CEA 1+1+1, триспецифическая одновалентная анти-CD28 (TGN1412), одновалентная анти-FAP и одновалентная анти-СЕА huIgG1 PG-LALA конструкция кросс-Fab, обмен VH/VL в анти-СЕА Kpocc-Fab-фрагменте (впадина), заряженные модификации в анти-FAP Fab-фрагменте (выступ) и в анти-CD28 фрагменте (выступ) (как на Фиг. 1Р). Молекула содержит аминокислотные последовательности из SEQ ID NO:87, 88, 388 и 389 (Р1АЕ4064).
1.2 Выработка биспецифических антигенсвязывающих молекул, нацеленных на CD28 и белок активации фибробластов (FAP) или карциноэмбриональный антиген (СЕА)
Экспрессией вышеуказанных молекул управляет химерный промотор MPSV или промотор CMV. Полиаденилирование обусловлено синтетической последовательностью сигнала поли-А, расположенной в 3' конце КП. Кроме того, каждый вектор содержит последовательность EBV OriP для аутосомальной репликации.
Для выработки конструкций С W, клетки HEK293-EBNA, которые росли в суспензии, котрансфицировали соответствующими экспрессионными векторами, используя в качестве трансфекционного реагента полиэтиленимин. Антитела и биспецифические антитела создавали с помощью временной трансфекции клеток НЕК293 EBNA. Клетки центрифугировали и заменяли среду предварительно нагретой средой CD СНО. Экспрессионные векторы смешивали в среде CD СНО, добавляли ПЭИ, перемешивали раствор на вортексе и инкубировали 10 минут при комнатной температуре. После этого клетки смешивали с раствором ДНК/ПЭИ, переносили во встряхиваемую колбу и инкубировали 3 часа при 37°С в инкубаторе с атмосферой 5% СО2. После инкубации добавляли среду Excell с добавками (культуры клеток млекопитающих для производства биопрепаратов, редакторы: Weichang Zhou, Anne Kantardjieff). Через одни сутки после трансфекции добавляли добавки (Feed) (культуры клеток млекопитающих для производства биопрепаратов, редакторы: Weichang Zhou, Anne Kantardjieff). Клеточные супернатанты собирали через 7 суток путем центрифугирования и последующей фильтрации (0,2 мкм фильтр) и очищали стандартными методами.
Конструкции А, В и X были изготовлены Evitria с использованием их собственной векторной системы с помощью стандартных (не на основе ПЦР) методик клонирования и использованием адаптированных к суспензии клеток СНО K1 (изначально полученных от АТСС и адаптированных для бессывороточного роста в суспензионной культуре в Evitria). Для выработки Evitria использовали их собственные не содержащую животных компонентов и не содержащую сыворотку среды (eviGrow и eviMake2) и их собственный трансфекционный реагент (eviFect). Супернатант собирали путем центрифугирования и последующей фильтрации (0,2 мкм фильтр) и очищали стандартными методами.
1.3 Очистка биспецифических антигенсвязывающих молекул, нацеленных на CD28 и белок активации фибробластов (FAP) или карциноэмбриональный антиген (СЕА)
Белки очищали из профильтрованных клеточных культуральных супернатантов согласно стандартным протоколам. Вкратце, Fc-содержащие белки очищали из клеточных культуральных супернатантов с помощью аффинной хроматографии с протеином А. Элюирование проводили при рН 3,0 с последующей немедленной нейтрализацией образца. Белок концентрировали и отделяли агрегированный белок от мономерного белка с помощью эксклюзионной хроматографии в 20 мМ гистидине, 140 мМ хлориде натрия, рН 6,0.
1.4 Аналитические данные по биспецифическим или триспецифическим антителам, нацеленным на CD28 и белок активации фибробластов (FAP) или карциноэмбриональный антиген (СЕА)
Концентрацию белка очищенных конструкций определяли путем измерения оптической плотности (ОП) на 280 нм, используя массовый коэффициент экстинкции, рассчитанный на основании аминокислотной последовательности, в соответствии с Расе, et al., Protein Science, 1995, 4, 2411-1423. Чистоту и молекулярную массу белков анализировали с помощью КЭ-ДСН в присутствии или отсутствие восстанавливающего агента, используя LabChipGXII (Perkin Elmer). Определение содержания агрегатов проводили с помощью ВЭЖХ-хроматографии при 25°С, используя аналитическую эксклюзионную колонку (TSKgel G3000 SW XL или UP-SW3000), уравновешенную в подвижном буфере (25 мМ K2HPO4, 125 мМ NaCl, 200 мМ L-аргинина моногидрохлорида, рН 6,7, или 200 мМ K2HPO4, 250 мМ KCl, рН 6,2, соответственно). Обобщенные данные по параметрам очистки всех молекул приведены в таблице 1.
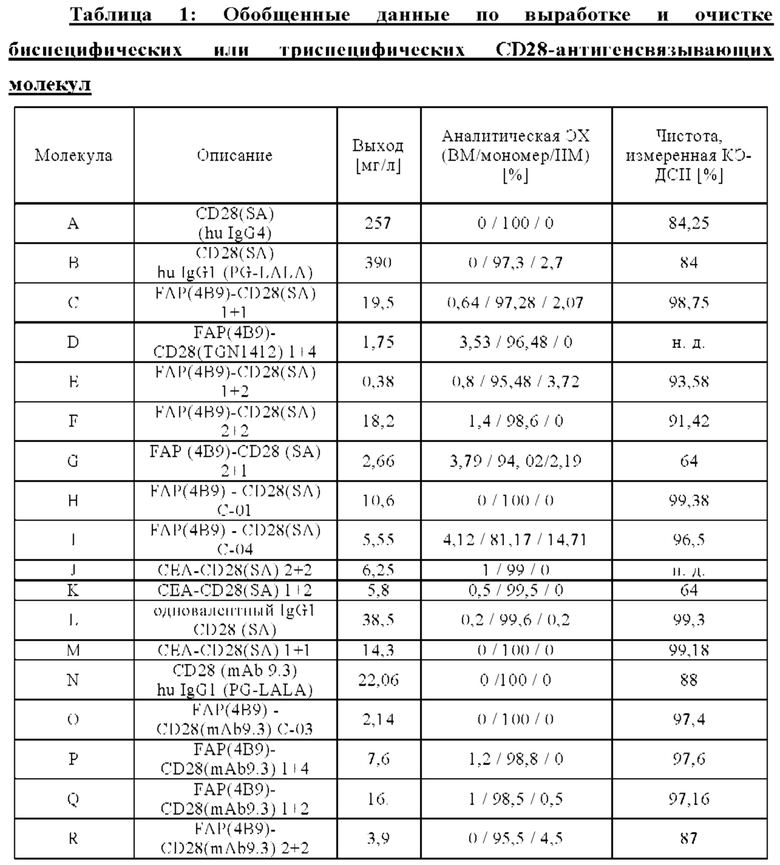
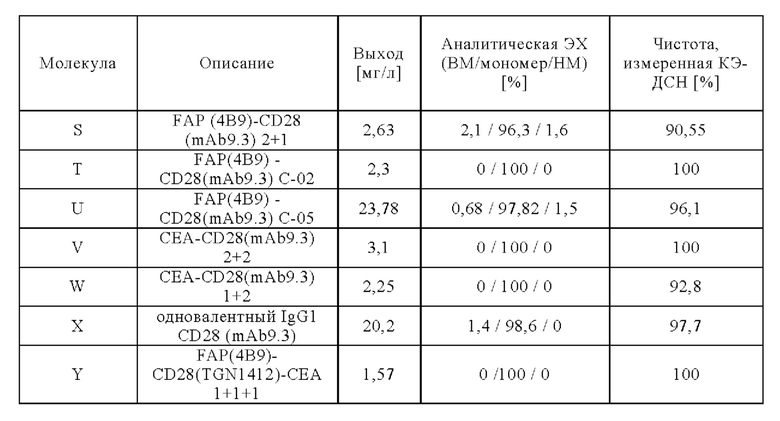
Пример 2
Связывание и кинетический анализ биспецифических антител или биспецифических антигенсвязывающих молекул, нацеленных на CD28 и белок активации фибробластов (FAP) или карциноэмбриональный антиген (СЕА)
2.1 Связывание биспецифических антител, нацеленных на CD28 и белок активации фибробластов (FAP) с FAP- или СЕА- и с CD28-экспрессирующими клетками
Связывание биспецифических молекул FAP-CD28 исследовали, используя экспрессирующие человеческий белок активации фибробластов (huFAP) клетки 3Т3-huFAP (клон 19). Линию клеток создавали путем трансфекции линии клеток мышиных эмбриональных фибробластов NIH/3T3 (АТСС CRL-1658) экспрессионным вектором pETR4921 для экспрессии huFAP в селективных условиях с 1,5 мкг/мл пуромицина. Связывание с CD28 человека исследовали с клетками СНО, экспрессирующими CD28 человека (родительская линия клеток CHO-k1 АТСС №CCL-61, модифицированная для стабильной сверхэкспрессии CD28 человека). Связывание с СЕАСАМ5 человека исследовали с СЕА-экспрессирующими клетками MKN45 (линия клеток рака желудка, DSMZ № АСС 409).
Чтобы оценить связывание, клетки собирали, подсчитывали, проверяли на жизнеспособность и ресуспендировали при 2,5Е5/мл в буфере FACS (eBioscience, кат. №00-4222-26). 5х104 клеток инкубировали в круглодонных 96-луночных планшетах в течение 2 ч при 4°С с возрастающими концентрациями FAP-нацеленных конструкций CD28 (1 пМ - 100 нМ). Потом клетки три раза промывали холодным буфером FACS, инкубировали еще 60 мин при 4°С с РЕ-конъюгированным, козьим античеловеческим РЕ (Jackson ImmunoReserach, кат. №109-116-098), промывали один раз холодным буфером FACS, центрифугировали и ресуспендировали в 100 μл буфера FACS. Для отслеживания неспецифических взаимодействий связывания между конструкциями и клетками в качестве отрицательного контроля был включен анти-DP47 IgG. Связывание оценивали методом проточной цитометрии с помощью FACS Fortessa (BD, программное обеспечение FACS Diva). Кривые связывания получали, используя GraphPadPrism6.
Молекулы FAP-CD28 были способны связываться с FAP человека, а также CD28 человека на клетках зависимым от концентрации образом (Фиг. 2Б и 2В для определенных примеров). Как и ожидалось, с анти-DP47 IgG связывание обнаружено не было, что свидетельствует о том, что обнаружение связывания обусловлено специфическим связыванием CD28 и FAP соответствующими нацеленными фрагментами.
Молекулы CEA-CD28 также были способны связываться с СЕА человека, а также CD28 человека на клетках.
2.2 Кинетический анализ биспецифических или триспецифических антител, нацеленных на CD28 и СЕА
Аффинность (KD) обоих связывающих фрагментов биспецифических или триспецифических антител, содержащих анти-СЕА (Medi-565) и анти-CD28, измеряли методом ППР, используя инструмент ProteOn XPR36 (Biorad) при 25°С с биотинилированным антигеном huCD28-Fc и биотинилированным Ни N(A2-B2)A-avi-His, иммобилизованными на чипе NLC путем захвата нейтравидина.
Для создания антигена на основе СЕАСАМ5, который содержит эпитоп для CEA(Medi-565), создавали химерный белок, состоящий из двух Ig-доменов СЕАСАМ1 и двух СЕАСАМ5. На основании последовательности СЕАСАМ1 второй и третий домены СЕАСАМ1 замещали доменами СЕАСАМ5 А2 и В2. С-концевой avi-тег и His-тег сливали для сайт-специфического биотинилирования и очистки. Полученный в результате белок был назван Hu N(A2-B2)A-avi-His (SEQ ID NO: 169).
Иммобилизация рекомбинантных антигенов (лиганд): Антигены разводили ФСБТ (10 мМ фосфата, 150 мМ хлорида натрия, рН 7,4, 0,005% Твин 20) до 10 мкг/мл, потом вводили при 30 мкл/минут с переменным временем контакта для обеспечения уровней иммобилизации около 400, 800 и 1600 единиц ответа (ЕО) в вертикальной ориентации. Введение аналитов: Для одноразовых измерений кинетики направление введения меняли на горизонтальную ориентацию, двукратные серийные разведения очищенного биспецифического нацеленного на СЕА анти-CD28 биспецифического антитела (диапазон переменной концентрации от 50 до 3,125 нМ) вводили одновременно при 50 мкл/мин вдоль отдельных каналов 1-5, со временем ассоциации 150 с и временем диссоциации 450 с. Буфер (ФСБТ) вводили вдоль шестого канала для обеспечения «параллельной» холостой пробы для сравнения. Константы скорости ассоциации (kon) и константы скорости диссоциации (koff) рассчитывали, используя простую один-к-одному модель связывания Лэнгмюра в программном обеспечении ProteOn Manager v3.1, путем одновременной аппроксимации сенсограмм ассоциации и диссоциации. Равновесную константу диссоциации (KD) рассчитывали, как соотношение koff/kon. Рассчитанные значения KD биспецифического антитела, содержащего один анти-CD28 антигенсвязывающий домен и один анти-СЕА антигенсвязывающий домен (молекула М), согласуются с измеренными значениями соответствующих моноспецифических конструкций. Кинетические и термодинамические данные обобщены в таблице 2 ниже.
Пример 3
Создание и изучение характеристик вариантов CD28 (SA) с отсутствием горячих точек и сниженной аффинностью
3.1 Удаление неспаренного остатка цистеина, остатков триптофана, сайта дезамидирования и создание вариантов CD28 (SA) со сниженной аффинностью
Как часть нашего подробного изучения характеристик связывающей молекулы проводили компьютерный анализ последовательностей вариабельного домена CD28(SA). Этот анализ выявил наличие неспаренного цистеина в области CDR2 VH (позиция 50, нумерация Kabat), остатков триптофана в CDR3 VH (позиция 100а, нумерация Kabat) и CDR1 VL (позиция 32, нумерация Kabat) и потенциального сайта дезамидирования аспарагина в CDR2 VH (позиция 56, нумерация Kabat). Хотя окисление триптофана является довольно медленным процессом и может быть предотвращено путем добавления восстанавливающих соединений, наличие неспаренных остатков цистеина в вариабельном домене антитела может быть критическим. Свободные остатки цистеина являются реакционноспособными и могут образовывать стабильные связи с другими неспаренными остатками цистеина других белков или компонентов клетки или среды. Как следствие, это может привести к получению гетерогенного и нестабильного продукта с неизвестными модификациями, которые являются потенциально иммуногенными и, следовательно, могут подвергать пациентов риску. Кроме того, дезамидирование аспарагина и последующее образование изоаспартата и сукцинимида может влиять как на in vitro стабильность, так и на in vivo биологические функции. Анализ кристаллической структуры родительской мышиной связывающей молекулы 5.11А выявил, что С50 не вовлечен в связывание с CD28 человека и, следовательно, может быть замещен схожей аминокислотой, такой как серии, не оказывая влияния на аффинность к CD28 (таблица 6, вариант 29). Однако оба остатка триптофана, а также аспарагин в позиции 50 расположены близко к или на поверхности связывания и, следовательно, замещение схожей аминокислотой может привести к снижению аффинности связывания. В этом примере нашей целью было, в частности, снижение аффинности CD28(SA) к CD28 человека по следующей причине: Аффинность CD28(SA) находится в диапазоне 12 нМ со временем полужизни связывания около 32 минут. Эта сильная аффинность может привести к эффекту поглощения в ткани, содержащей большие количества CD28-экспрессирующих клеток, такой как кровь и лимфа, при внутривенном введении пациентам, как следствие сайт-специфическое нацеливание соединения посредством нацеливающих компонентов FAP и/или CEA может быть снижено и эффективность конструкции может быть уменьшена. Чтобы свести к минимуму такой эффект, создавали несколько вариантов VH и VL для снижения аффинности в разной степени (Фиг. 3А и 3В). Помимо ранее упомянутых позиций, которые представляют потенциальные горячие точки стабильности, дополнительные остатки, прямо или непрямо участвующие в связывании с CD28 человека, замещали оригинальной аминокислотой мышиной зародышевой линии или сходной аминокислотой, дополнительно CDR VL и VH CD28(SA) также прививали в соответствующие каркасные последовательности трастузумаба (Фиг. 3Б и 3Г). Потом несколько комбинаций вариантов VH и VL экспрессировали в виде одновалентных анти-CD28 IgG-подобных конструкций с одним плечом и изучали характеристики связывания методом ППР.
3.2 Анализ констант скорости диссоциации (koff) уменьшенных анти-CD28 вариантов с одним плечом методом ППР
Для получения характеристик анти-CD28 связывающих вариантов на первом этапе все связывающие молекулы экспрессировали в виде одновалентных IgG-подобных конструкций с одним плечом (Фиг. 4А). Это формат был выбран для изучения характеристик связывания с CD28 в модели 1:1. Через 5 суток после трансфекции в клетки HEK собирали супернатант и определяли титр экспрессируемых конструкций.
Скорость диссоциации анти-CD28 связывающих вариантов определяли методом поверхностного плазмонного резонанса (ППР), используя инструмент ProteOn XPR36 (Biorad) при 25°С с биотинилированным антигеном huCD28-Fc, иммобилизованным на чипах NLC путем захвата нейтравидина. Для иммобилизации рекомбинантного антигена (лиганда) huCD28-Fc разводили ФСБТ (фосфатно-солевой буфер с Твин 20, состоящий из 10 мМ фосфата, 150 мМ хлорида натрия, рН 7,4, 0,005% Твин 20), до концентрации в диапазоне от 100 до 500 нМ, потом вводили при 25 мкл/минута с переменным временем контакта. Это приводило к уровням иммобилизации от 1000 до 3000 единиц ответа (ЕО) в вертикальной ориентации.
Для одноразовых измерений кинетики направление введения меняли на горизонтальную ориентацию. На основании титра полученных супернатантов одновалентные IgG с одним плечом разводили в ФСБТ для получения двукратных серийных разведений в диапазоне от 100 нМ до 6,25 нМ. Введение проводили одновременно при 50 мкл/мин вдоль отдельных каналов 15, со временем ассоциации 120 с и временем диссоциации 300 с. Буфер (ФСБТ) вводили вдоль шестого канала для обеспечения «параллельной» холостой пробы для сравнения. Поскольку взаимодействие связывания измеряли с одновалентными IgG с одним плечом из супернатанта без очистки и биохимического анализа, для дальнейших заключений использовали только значения скорости диссоциации взаимодействия белок:белок. Значения скорости диссоциации рассчитывали, используя простую один-к-одному модель связывания Лэнгмюра в программном обеспечении ProteOn Manager v3.1, путем аппроксимации сенсограмм диссоциации. Значения констант скорости диссоциации (koff) всех клонов обобщены в таблице 3. Сравнение полученных вариантов позволило выявить значения koff с 30-кратным снижением по сравнению с родительской последовательностью.
Связывание с CD28 человека исследовали с клетками СНО, экспрессирующими CD28 человека (родительская линия клеток CHO-k1 АТСС №CCL-61). Этот анализ связывания описан в примере 4 ниже. Одновалентные IgG-подобные CD28 вариантные конструкции с одним плечом демонстрировали разницу в связывании, как можно видеть на Фиг. 4А-4В.
3.3 Получение и кинетический анализ биспецифических нацеленных на FAP анти-CD28 вариантов по аффинности
На основании анализа скорости диссоциации и исследования связывания на CD28-экспрессирующих клетках отобрали несколько комбинаций анти-CD28 вариантов VH и VL с разной интенсивностью связывания и экспрессировали их в виде нацеленных на FAP биспецифических huIgGl PG-LALA кросс-Fab молекул (комбинации SEQ ID NO: смотрите в таблице 4). Полученные в результате конструкции в формате 1+1 (Фиг. 4Б) очищали и проводили биохимический анализ (таблица 5).
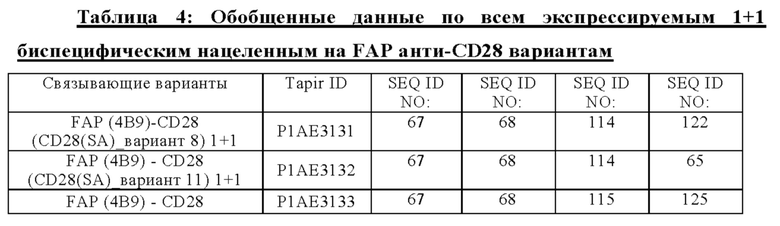
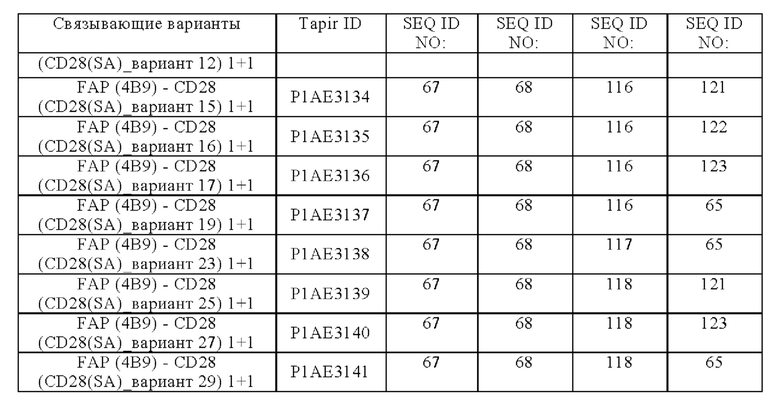
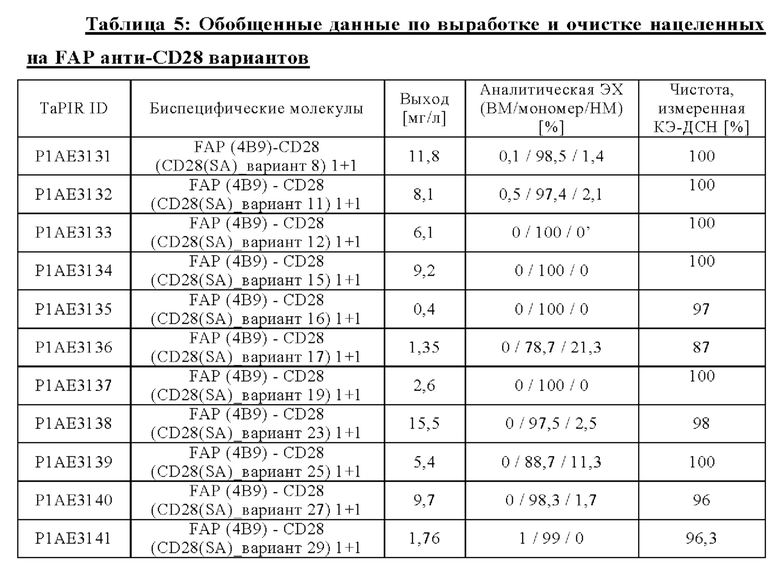
Аффинность (KD) полученных биспецифических антигенсвязывающих молекул к CD28 измеряли методом ППР, используя инструмент ProteOn XPR36 (Biorad) при 25°С с биотинилированным антигеном huCD28-Fc, иммобилизованным на чипах NLC путем захвата нейтравидина. Иммобилизация рекомбинантных антигенов (лиганд): Антиген разводили ФСБТ (10 мМ фосфата, 150 мМ хлорида натрия, рН 7,4, 0,005% Твин 20) до 10 мкг/мл, потом вводили при 30 мкл/минут с переменным временем контакта для обеспечения уровней иммобилизации около 200, 400 или 800 единиц ответа (ЕО) в вертикальной ориентации. Введение аналитов: Для одноразовых измерений кинетики направление введения меняли на горизонтальную ориентацию, двукратные серийные разведения очищенных биспецифических нацеленных на FAP анти-CD28 вариантов по аффинности (диапазон переменной концентрации от 50 до 3,125 нМ) вводили одновременно при 50 мкл/мин вдоль отдельных каналов 1-5, со временем ассоциации 150 с и временем диссоциации 450 с. Буфер (ФСБТ) вводили вдоль шестого канала для обеспечения «параллельной» холостой пробы для сравнения. Константы скорости ассоциации (kon) и константы скорости диссоциации (koff) рассчитывали, используя простую один-к-одному модель связывания Лэнгмюра в программном обеспечении ProteOn Manager v3.1, путем одновременной аппроксимации сенсограмм ассоциации и диссоциации. Равновесную константу диссоциации (KD) рассчитывали как соотношение koff/kon. Анализируемые клоны демонстрировали значения KD в широком диапазоне (от 1 до 25 нМ). Кинетические и термодинамические данные обобщены в таблице 6.
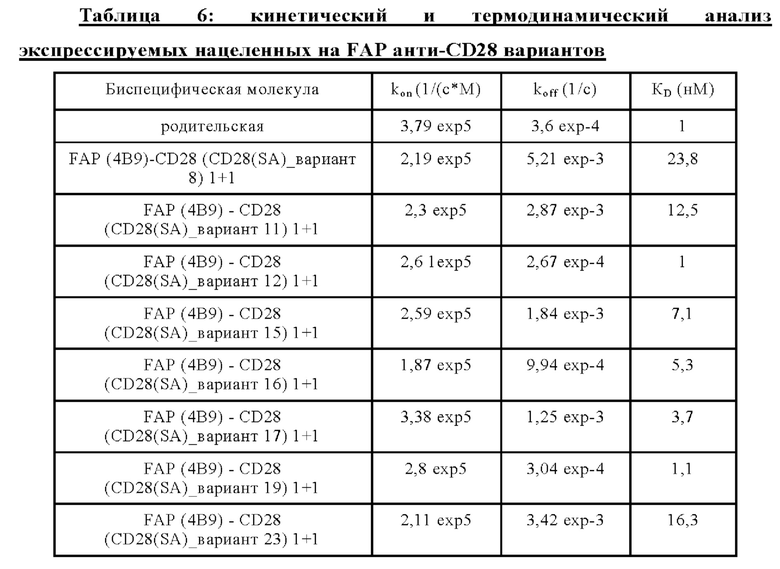
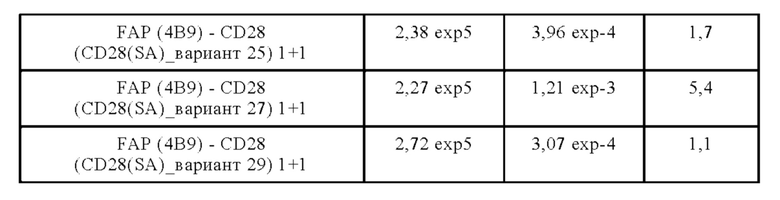
Пример 4
Связывание одновалентных CD28 агонистических IgG и нацеленных на FAP агонистических антител к CD28 с CD28-экспрессирующими и FAP-экспрессирующими клетками
Связывание с CD28 человека исследовали с клетками СНО, экспрессирующими CD28 человека (родительская линия клеток CHO-k1 АТСС №CCL-61, модифицированная для стабильной сверхэкспрессии CD28 человека). Чтобы оценить связывание, клетки собирали, подсчитывали, проверяли на жизнеспособность и ресуспендировали при 2,5х105/мл в буфере FACS (eBioscience, кат.№00-4222-26). 5х104 клеток инкубировали в круглодонных 96-луночных планшетах в течение 2 ч при 4°С с возрастающими концентрациями, связывающих CD28 молекул (1 пМ - 100 нМ). Потом клетки три раза промывали холодным буфером FACS, инкубировали еще 60 мин при 4°С с РЕ-конъюгированным, козьим античеловеческим РЕ (Jackson ImmunoReserach, кат. №109-116-098), промывали один раз холодным буфером FACS, центрифугировали и ресуспендировали в 100 мкл буфера FACS. Для отслеживания неспецифических взаимодействий связывания между конструкциями и клетками в качестве отрицательного контроля был включен анти-DP47 IgG. Связывание оценивали методом проточной цитометрии с помощью FACS Fortessa (BD, программное обеспечение FACS Diva). Кривые связывания получали, используя GraphPadPrism6.
Одновалентные IgG-подобные CD28 вариантные конструкции с одним плечом демонстрировали разницу в связывании, как можно видеть на Фиг. 4А-4В. Кроме того, определяли связывание биспецифических нацеленных на FAP анти-CD28 антител в формате 1+1 с клетками СНО, экспрессирующими CD28 человека. Значения KD для разных 1+1 конструкций с отобранными вариантами CD28 приведены в таблице 7 ниже или на соответствующих графиках на Фиг. 4Г и 4Д.
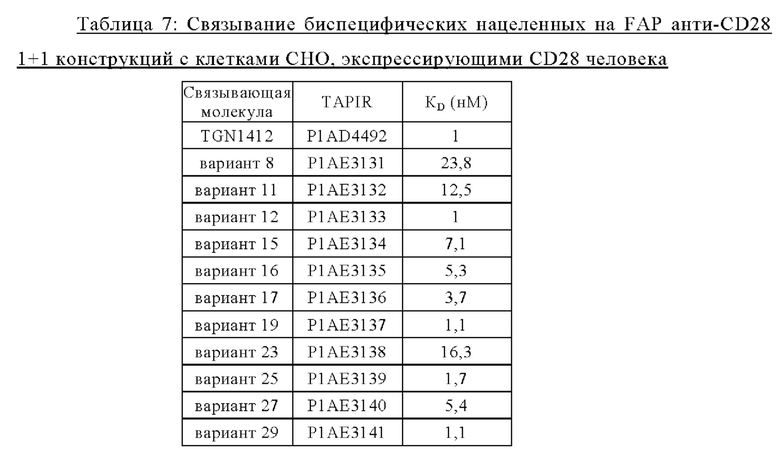
Связывание биспецифических нацеленных на FAP анти-CD28 антител в формате 1+1 с FAP-экспрессирующими клетками 3Т3-huFAP (клон 19) также определяли, как описано в примере 2.1 и показано на соответствующих графиках на Фиг. 4Е и 4Ж.
Пример 5
Связывание нацеленных на СЕА агонистических антител к CD28 с CD28-экспрессирующими клетками
Связывание с CD28 человека исследовали в клетках СНО, экспрессирующих CD28 человека (связывание оценивали методом проточной цитометрии с помощью FACS Fortessa (BD, программное обеспечение FACS Diva)), как описано в примере 4. Кривые связывания получали, используя GraphPadPrism6. Кривые связывания для разных 1+1 конструкций с отобранными вариантами CD28 приведены на Фиг. 16.
Пример 6
In vitro функциональные характеристики нацеливания на CD28 и белок активации фибробластов (FAP) или карциноэмбриональный антиген (СЕА)
Проводили несколько клеточных in vitro анализов с первичными человеческими МКПК, чтобы оценить активность CD28(SA) и биспецифических нацеленных на FAP CD28-антигенсвязывающих молекул в присутствии или отсутствие сигналов ТКР, обеспечиваемых Т-клеточными биспецифическими (ТСВ) антителами. Пролиферацию Т-клеток, секрецию цитокинов и уничтожение опухолевых клеток, определяемые с помощью проточной цитометрии, ELISA цитокинов и визуализации живых клеток, получали как результаты измерений.
1. Активность оригинального суперагонистического CD28(SA) IgG4 оценивали, используя ранее описанную систему предварительного культивирования при высокой плотности для восстановления реактивности полученных из периферической крови Т-клеток на CD28-опосредованный суперагонизм (Romer et al., 2011).
2. Функциональность нацеленных молекул CD28 в отсутствие сигналов ТКР оценивали в анализе совместной культуры первичных человеческих МКПК, при этом нацеленные на FAP или СЕА молекулы CD28 были перекрестно связаны за счет одновременного связывания с CD28 человека на Т-клетках и FAP человека, экспрессируемым на клетках 3Т3-huFAP (родительская линия клеток АТСС № CCL-92, модифицированная для стабильной сверхэкспрессии FAP человека) или на MCSP- и FAP-экспрессирующих клетках меланомы MV3, СЕА-экспрессирующих клетках рака желудка MKN45, соответственно.
3. Функциональность нацеленных на FAP молекул CD28 в присутствии сигналов ТКР оценивали, как описано выше, с дополнительным присутствием молекулы ТСВ, перекрестно связанной за счет одновременного связывания с CD3 на Т-клетках и СЕА человека на клетках рака желудка MKN45, клетках рака толстой кишки Lovo, клетках рака толстой кишки НТ-29, или MCSP, экспрессируемым на клетках меланомы MV3.
Выделение МКПК
Мононуклеарные клетки периферической крови (МКПК) получали с помощью центрифугирования в градиенте плотности из обогащенных лимфоцитарных препаратов гепаринизированной крови, полученных из лейкоцитаной пленки (Blutspende Zurich). 25 мл крови (разведенной 1:2 в ФСБ) наносили слоем на 15 мл реагента lymphoprep (STEMCELL technologies, кат. №07851) и центрифугировали при комнатной температуре в течение 25 мин при 845xg без перерыва. Содержащую МКПК межфазную поверхность собирали в 50 мл пробирки с помощью 10 мл пипетки. Клетки промывали ФСБ и центрифугировали 5 мин при 611xg. Супернатант сливали, осадок ресуспендировали в 50 мл ФСБ и центрифугировали 5 мин при 304xg. Этап промывки повторяли и центрифугировали при 171xg. Клетки ресуспендировали в RPMI 1640 Glutamax (содержащем 5% человеческую сыворотку, пируват натрия, NEAA, 50 μΜ 2-меркаптоэтанол, пенициллин/стрептомицин) и обрабатывали для дальнейшего функционального анализа согласно протоколу соответствующего анализа.
Предварительное культивирование МКПК при высокой плотности и in vitro оценка активации Т-клеток суперагонистом CD28 CD28(SA)
Для восстановления реактивности человеческих Т-клеток на TGN1412-опосредованный суперагонизм CD28 МКПК предварительно культивировали при высокой плотности (ВП) ( et al, 2011) перед оценкой эффектов суперагонистических антител к CD28. Вкратце, МКПК доводили до 1Е7 клеток/мл в полной среде (RPMI 1640 Glutamax, 5% человеческая сыворотка, пируват натрия, NEAA, 50 мкМ 2-меркаптоэтанол, пенициллин/стрептомицин) и культивировали при 1,5 мл/лунка в 24-луночном планшете в течение 48 часов при 37°С, 5% СО2. Потом клетки повторно собирали, промывали в полной среде, центрифугировали при 550xg в течение 5 мин и доводили до желаемой плотности клеток, необходимой для изучения функциональных характеристик. Чтобы оценить пролиферацию Т-клеток, МКПК метили CFSE и измеряли разведение CFSE как показатель пролиферации Т-клеток через 5 суток после стимуляции. Вкратце, клетки доводили до 2х107/мл в ФСБ и метили 2,5 μΜ красителя для оценки пролиферации CFSE (LifeTechnologies, кат. №65-0850-84) в течение 6 мин при 37°С, 5% СО2. Клетки один раз промывали в полной среде с последующими 2 этапами в ФСБ. Для стимуляции TGN1412 МКПК доводили до 2х106/мл в полной среде и распределяли 1х105 в каждую лунку плоскодонного 96-луночного планшета и стимулировали возрастающими концентрациями TGN1412 (от 0,0002 нМ до 10 нМ, три повтора). Разведение CFSE оценивали методом проточной цитометрии. Вкратце, клетки центрифугировали при 550xg в течение 5 мин и промывали ФСБ. Разведение CFSE оценивали методом проточной цитометрии. Вкратце, клетки центрифугировали при 550xg в течение 5 мин и промывали ФСБ. Поверхностное окрашивание в отношении CD8 (BV711 античеловеческий CD8a, BioLegend №301044), CD4 (РЕ-Су7 античеловеческий CD4, BioLegend №344612) проводили в соответствии с указаниями поставщика. Потом клетки дважды промывали 150 мкл/лунка ФСБ и ресуспендировали в 200 мкл/лунка буфера FACS и анализировали, используя BD FACS Fortessa. Секрецию цитокинов измеряли на 5 сутки после активации с помощью ELISA цитокинов (huTNFα, DuoSet №DY210-05 и huIFNγ, DuoSet № DY285-05) или многоканального анализа цитокинов (17-канальный анализ цитокинов человека, Bio-Rad № M5000031YV) из культуральных супернатантов.
et al, 2011) перед оценкой эффектов суперагонистических антител к CD28. Вкратце, МКПК доводили до 1Е7 клеток/мл в полной среде (RPMI 1640 Glutamax, 5% человеческая сыворотка, пируват натрия, NEAA, 50 мкМ 2-меркаптоэтанол, пенициллин/стрептомицин) и культивировали при 1,5 мл/лунка в 24-луночном планшете в течение 48 часов при 37°С, 5% СО2. Потом клетки повторно собирали, промывали в полной среде, центрифугировали при 550xg в течение 5 мин и доводили до желаемой плотности клеток, необходимой для изучения функциональных характеристик. Чтобы оценить пролиферацию Т-клеток, МКПК метили CFSE и измеряли разведение CFSE как показатель пролиферации Т-клеток через 5 суток после стимуляции. Вкратце, клетки доводили до 2х107/мл в ФСБ и метили 2,5 μΜ красителя для оценки пролиферации CFSE (LifeTechnologies, кат. №65-0850-84) в течение 6 мин при 37°С, 5% СО2. Клетки один раз промывали в полной среде с последующими 2 этапами в ФСБ. Для стимуляции TGN1412 МКПК доводили до 2х106/мл в полной среде и распределяли 1х105 в каждую лунку плоскодонного 96-луночного планшета и стимулировали возрастающими концентрациями TGN1412 (от 0,0002 нМ до 10 нМ, три повтора). Разведение CFSE оценивали методом проточной цитометрии. Вкратце, клетки центрифугировали при 550xg в течение 5 мин и промывали ФСБ. Разведение CFSE оценивали методом проточной цитометрии. Вкратце, клетки центрифугировали при 550xg в течение 5 мин и промывали ФСБ. Поверхностное окрашивание в отношении CD8 (BV711 античеловеческий CD8a, BioLegend №301044), CD4 (РЕ-Су7 античеловеческий CD4, BioLegend №344612) проводили в соответствии с указаниями поставщика. Потом клетки дважды промывали 150 мкл/лунка ФСБ и ресуспендировали в 200 мкл/лунка буфера FACS и анализировали, используя BD FACS Fortessa. Секрецию цитокинов измеряли на 5 сутки после активации с помощью ELISA цитокинов (huTNFα, DuoSet №DY210-05 и huIFNγ, DuoSet № DY285-05) или многоканального анализа цитокинов (17-канальный анализ цитокинов человека, Bio-Rad № M5000031YV) из культуральных супернатантов.
In vitro оценка пролиферации Т-клеток и секреции цитокинов биспецифическими нацеленными на FAP CD28-антигенсвязывающими молекулами в отсутствие и присутствии сигналов ТСВ
Пан-Т-клетки использовали в качестве эффекторных клеток и выделяли из МКПК с помощью MACS, используя набор для выделения пан-Т-клеток (Miltenyi Biotec) в соответствии с инструкциями производителя.
Для измерения активации Т-клеток биспецифическими FAP-CD28-антигенсвязывающими молекулами в отсутствие ТСВ, меченные CFSE пан-Т-клетки культивировали совместно с 3х104/лунка клеток 3Т3-huFAP или родительских клеток 3Т3 с отсутствием экспрессии FAP (3Т3-ДТ), которые за сутки до этого были высеяны в плоскодонные 96-луночные планшеты. Биспецифические FAP-CD28-антигенсвязывающие молекулы добавляли в возрастающих концентрациях (0,0002 нМ 10 нМ, три повтора).
Для измерения пролиферации Т-клеток в присутствии сигнала ТСВ, меченные CFSE пан-Т-клетки инкубировали с 3х104 экспрессирующих FAP и MCSP клеток/лунка MV3, которые за сутки до этого были высеяны в плоскодонные 96-луночные планшеты, возрастающими концентрациями биспецифических FAP-CD28-антигенсвязывающих молекул (0,0002 нМ 10 нМ, три повтора) и фиксированной концентрацией MCSP-TCB (5 пМ, P1AD2189). В качестве контроля были включены лунки, содержащие только ТСВ.
Разведение CFSE оценивали методом проточной цитометрии, а секрецию цитокинов измеряли на 5 сутки после активации с помощью ELISA цитокинов (huTNFα, DuoSet №DY210-05 и huIFNγ, DuoSet №DY285-05) или многоканального анализа цитокинов (17-канальный анализ цитокинов человека, Bio-Rad №M5000031YV) из культуральных супернатантов.
Получение анти-MCSP/анти-CD3 биспецифического антитела (MCSP-TCB), используемого в этом эксперименте, описано в WO 2014/131712 А1.
Для суперагонизма CD28(SA) необходимо перекрестное связывание FcγRIIb
Предварительное культивирование МКПК при высокой плотности восстанавливает суперагонизм CD28(SA)
Чтобы понять механизм действия CD28(SA), мы подтверждали, что предварительное культивирование МКПК при высокой плотности (ВП) согласно ранее описанному протоколу восстанавливает способность полученных из МКПК Т-клеток отвечать на TGN1412-опосредованный суперагонизм CD28 (Romer et al., 2011). Как изображено на Фиг. 5А и 5Б, CD28(SA) IgG4 (Р1АЕ1975) индуцирует пролиферацию Т-клеток МКПК (Фиг. 5А) и выработку цитокинов (Фиг. 5Б) зависимым от концентрации образом через 5 суток после стимуляции только в МКПК, которые перед этим культивировали при ВП, тогда как свежие МКПК остались нереактивными. Мы сделали заключение, что мы можем воспроизвести ранее опубликованный протокол для восстановления реактивности Т-клеток на CD28(SA) in vitro (Romer et al., 2011).
Для суперагонистической активности CD28(SA) необходимо перекрестное связывание посредством via FcγRIIb Блокирование FcγRIIb снижает функциональность CD28(SA)
В ранее опубликованных литературных данных указано, что TGN1412 потенциально зависит от перекрестного связывания FcγRIIb. Чтобы понять взаимосвязь между предварительным культивированием МКПК при ВП и Fc-зависимостью функциональности CD28(SA), оценивали уровни экспрессии FcγRIIb на МКПК методом проточной цитометрии до и после предварительного культивирования при ВП. Как изображено на Фиг. 5В, экспрессия FcγRIIb отсутствовала в свежих моноцитах МКПК, тогда как 96,8% моноцитов экспрессировали FcγRIIb через 2 суток после предварительного культивирования при ВП. Опосредованное антителом блокирование FcγRIIb в последующем анализе пролиферации Т-клеток полностью устраняло пролиферацию Т-клеток после стимуляции CD28(SA) по данным измерения через 5 суток в культуре (Фиг. 5Г). В альтернативном подходе вариант CD28(SA) с Fc-сайленсингом, который несет мутацию P329G-LALA (CD28(SA) IgG1 PG-LALA: P1AD9289), не демонстрировал суперагонистической функции (Фиг. 6А). Эти данные подтверждают, что CD28(SA)-опосредованный суперагонизм CD28 зависит от перекрестного связывания посредством FcγRIIb.
Добавление нацеленного на опухоль фрагмента для нацеливания FAP на Fc-сайленсинг CD28(SA) восстанавливает суперагонизм, который затем зависит от наличия опухолевой мишени
Учитывая, что суперагонизм CD28, обусловленный TGN1412, зависит от перекрестного связывания FcγRIIb, мы предположили, что FcR-зависимость можно перенаправить на опухоли путем внесения (i) мутации P329G-LALA для Fc-сайленсинга и (ii) нацеливания фрагмента, который перекрестно связывается с экспрессируемым на поверхности опухолевым антигеном. Чтобы проверить эту гипотезу добавляли нацеленный на FAP фрагмент в С-концевое слияние TGN1412 с Fc-сайленсингом (FAP-CD28 1+2 SA: P1AD9011). Поскольку для этого подхода не было необходимо перекрестное связывание FcR, МКПК не подвергали предварительному культивированию при ВП. Вместо этого свежие МКПК культивировали совместно с 3Т3-huFAP или 3Т3-ДТ в течение 5 суток в присутствии возрастающих концентраций FAP-CD28 (P1AD9011) и оценивали пролиферацию Т-клеток по разведению CFSE методом проточной цитометрии. Как показано на Фиг. 6Б, внесение FAP-связывающего фрагмента делает возможной пролиферацию Т-клеток исключительно в присутствии FAP. Мы сделали заключение, что суперагонизм можно избирательно нацеливать на опухолевые антигены путем Fc-сайленсинга и добавления нацеленного на опухоль фрагмента.
Стандартные агонистические антитела к CD28 (клон 9.3) не проявляют суперагонистическое поведение в нацеленных на опухоль биспецифических форматах.
В литературе описаны два агонистических антитела к CD28: суперагонистические антитела к CD28, такие как TGN1412, способны автономно активировать Т-клетки без необходимости в дополнительных сигналах, обеспечиваемых ТКР. Эти антитела называют суперагонистами, потому что они превосходят по функциональности природные агонистические лиганды CD28, CD80 и CD86, которые сильно зависят от наличия сигнала ТКР для повышения Т-клеточной функции. В отличие от суперагонистических антител, таких как TGN1412, стандартные агонистические антитела, такие как клон mab 9.3, не способны активировать Т-клетки автономно, но, как и в случае природных лигандов CD28, для повышения активности Т-клеток необходим дополнительный сигнал ТКР. Чтобы оценить эффект нацеливания агонистов CD28 на опухолевые антигены более подробно, мы создали дополнительные молекулы FAP-CD28: (i) суперагонистическую (SA) молекулу с 2 CD28-связывающими фрагментами (TGN1412) и 2 FAP-связывающими фрагментами = формат 2+2 SA (P1AD4493), (ii) стандартный агонист (СА) с 2 CD28-связывающими фрагментами (клон 9.3) и 1 или 2 FAP-связывающими фрагментами, соответственно: 2+2 CA (P1AD8968), 1+2 CA {P1AD8962). Свежие МКПК культивировали совместно с 3Т3-huFAP или 3Т3-ДТ в течение 5 суток в присутствии возрастающих концентраций нацеленных на FAP молекул и оценивали пролиферацию Т-клеток по разведению CFSE методом проточной цитометрии. Как изображено на Фиг. 7А-7Г, только суперагонистические связывающие молекулы были способны активировать Т-клетки. Кроме того, активация Т-клеток посредством описанных суперагонистических конструкция сильно зависит от наличия FAP (Фиг. 7Б), что продемонстрировано отсутствием активации Т-клеток в отсутствие FAP (Фиг. 7Г). С этими данными также согласуется то, что секрецию цитокинов наблюдали только для конструкций, содержащих только суперагонистические CD28(SA) антитела, но не стандартное агонистическое антитело 9.3 (Фиг. 7Д). Мы сделали заключение, что только суперагонистические антитела к CD28 вызывают автономную активацию Т-клеток в биспецифических нацеленных на опухоль форматах антител, тогда как те же самые форматы со стандартными связывающими молекулами 9.3 не являются суперагонистическими.
Пример 7
In vitro оценка уничтожения опухолевых клеток нацеленными на опухоль молекулами CD28 в отсутствие или присутствии ТСВ
Чтобы оценить способность биспецифических FAP-CD28- или CEA-CD28-антигенсвязывающих молекул обеспечивать уничтожение опухолевых клеток или поддерживать ТСВ-опосредованное уничтожение опухолевых клеток, очищенные пан-Т-клетки служили в качестве эффекторных клеток, а экспрессирующие RFP клетки MV3 и клетки MKN45, соответственно, служили в качестве опухолевых мишеней.
Чтобы оценить уничтожение опухолевых клеток MV3, 5000 клеток-мишеней MV3, высеянных на предыдущие сутки, культивировали совместно с 1x105 пан-Т-клеток на лунку в плоскодонных 96-луночных планшетах (Э:М 20:1) в присутствии 5 пМ MCSP-TCB (P1AD2189), одного или в комбинации с 10 нМ биспецифической FAP-CD28 антигенсвязывающей молекулы. Чтобы оценить уничтожение опухолевых клеток MV3, 5000 клеток-мишеней MV3, высеянных на предыдущие сутки, культивировали совместно с 1х105 пан-Т-клеток на лунку в плоскодонных 96-луночных планшетах (Э:М 20:1) в присутствии 2 нМ FAP-CD28. Чтобы оценить уничтожение опухолевых клеток MKN45, 5000 MKN45, высеянных на предыдущие сутки, культивировали совместно с 1х105 пан-Т-клеток на лунку в плоскодонных 96-луночных планшетах в присутствии 2 нМ CEA-CD28. Уничтожение клеток-мишеней отслеживали в течение 90 часов, используя систему для визуализации живых клеток IncuCyte (Essen Biosciences), делая по 4 снимка на лунку каждые 3 часа. Число RFP+объектов на изображение (по оценке с помощью программного обеспечения IncuCyte ZOOM, Essen Biosciences) в течение времени служило показателем гибели клеток-мишеней. Опосредованное антителом уничтожение клеток-мишеней отличали от спонтанной гибели клеток-мишеней путем отслеживания числа клеток-мишеней в присутствии одних эффекторных Т-клеток в течение времени (= контроль базовой линии). Уничтожение рассчитывали как 100 - х, где x представляет % мишеней относительно контроля базовой линии. Статистический анализ проводили, используя t-критерий Стьюдента, сравнивая площади под кривыми (ППК) % уничтожения в течение времени.
FAP-CD28 индуцирует уничтожение клеток-мишеней в формате 1+2. но только с суперагонистическими связывающими CD28 молекулами, но не со стандартными агонистическими связывающими CD28 молекулами
Оценивали способность молекул FAP-CD28 индуцировать уничтожение опухолевых клеток. Как изображено на Фиг. 8А-8Г, совместное культивирование полученных из МКПК Т-клеток с экспрессирующими FAP клетками меланомы MV3 в присутствии FAP-CD28 в течение 90 часов приводило к уничтожению клеток MV3 исключительно FAP CD28(SA) в формате 1+2 (P1AD9011) и было сравнимо с индукцией уничтожения, обеспечиваемой FAP-нацеленными ТСВ (7). С FAP-CD28(SA) в формате 2+2 (P1AD4493), а также с FAP-CD28 со стандартным агонистическим антителом к CD28 9.3 (P1AD8968 & P1AD8962) уничтожение не наблюдали. Мы сделали заключение, что помимо пролиферации Т-клеток и секреции цитокинов, FAP-CD28 в формате 1+2 с суперагонистическими связывающими молекулами также вызывает уничтожение клеток-мишеней, сравнимое с ТСВ.
CEA-CD28 индуцирует уничтожение клеток-мишеней в формате 1+2 и 2+2, но только с суперагонистическими антителами, но не со стандартными агонистическими антителами к CD28
В альтернативном подходе мы использовали нацеленные на СЕА агонистические молекулы CD28 в форматах 2+2 SA (Р1АЕ1195), 1+2 SA (Р1АЕ1194), 2+2 СА (Р1АЕ1193), и 2+1 СА (Р1АЕ1192) для оценки их способности индуцировать уничтожение клеток-мишеней. Т-клетки МКПУ культивировали совместно с экспрессирующими СЕА клетками MKN45 в присутствии CEA-CD28 в вышеуказанных форматах в течение 90 ч. Оба формата, содержащие суперагонистические связывающие CD28 были способны индуцировать уничтожение экспрессирующих СЕА клеток MKN45 (Фиг. 9А и 9Б). Мы предполагаем, что расхождения между способностью FAP-CD28(SA) 2+2 и CEA-CD28 (SA) 2+2 уничтожать их соответствующие клетки-мишени связано с расхождением уровней экспрессии мишеней в клетках MKN45 и MV3. Точнее, лабораторные данные подтвердили, что уровни экспрессии FAP клеток MV3 являются в 10х меньшими, чем уровни экспрессии СЕА клеток MKN45. Таким образом, в клетках MV3 сайты связывания опухолевой мишени могут быть ограничены и для уничтожения клеток MV3 необходима эффективная занятость FAP относительно CD28, которая является более преимущественной в формате 1+2 (т.е. 1 сайт связывания FAP перекрестно связывает 2 сайта связывания CD28) по сравнению с 2+2 (т.е. 2 сайта связывания FAP необходимы для перекрестного связывания 2 сайтов связывания CD28).
Суперагонизм CD28 связывающими молекулами TGN1412 зависит от многовалентности связывающей CD28 молекулы одновалентные связывающие молекулы не являются суперагонистическими
Чтобы дополнительно исследовать природу суперагонизма CD28, мы оценивали, будут ли одновалентные связывающие молекулы CD28 TGN1412 демонстрировать суперагонистическое поведение в нацеленных на опухоль биспецифических форматах. Т-клетки МКПК культивировали совместно с клетками ЗТ3-huFAP и инкубировали с возрастающими концентрациями FAP-CD28 1+2 SA с двухвалентностью к CD28 (P1AD9011) и FAP-CD28 1+1 SA с одновалентностью к CD28 (P1AD4492). Как показано на Фиг. 10А, FAP-CD28 с одновалентным связыванием CD28 (Ρ1AD4492) не был способен индуцировать пролиферацию Т-клеток в отличие от двухвалентной конструкции CD28 (P1AD9011). Соответственно, повышение маркеров активации Т-клеток CD69 и CD25 наблюдали только при двухвалентности к CD28 (Фиг. 10Б и 10 В, соответственно). В заключение, TGN1412-опосредованный суперагонизм не только зависит от перекрестного связывания посредством Fc-рецепторов, но также для него необходима многовалентность связывающей CD28 молекулы.
В заключение, можно установить, что суперагонизм CD28 можно специфически нацеливать на опухолевые антигены путем Fc-сайленсинга и внесения антигенсвязывающего домена, способного к специфическому связыванию с опухолеассоциированным антигеном. Кроме того, нацеленные на опухоль биспецифические антитела являются суперагонистическими, только когда содержат связывающие молекулы на основе CD28(SA), но не когда они содержат стандартные агонистические связывающие молекулы (клон 9.3). Кроме того, для суперагонизма необходима многовалентность связывающей молекулы CD28(SA), а одновалентное связывание CD28(SA) в биспецифических конструкциях устраняет суперагонистическую активацию Т-клеток.
FAP-CD28 поддерживает ТСВ-опосредованное уничтожение клеток-мишеней и требует одновалентности связывающей CD28 молекулы для обеспечения зависимости от опухолевой мишени
Сигнализация CD28 описана как усиливающая опосредованные Т-клеточным рецептором ответы Т-клеток. Следовательно, Т-клеточные биспецифические антитела (ТСВ) являются перспективными партнерами для комбинации для агонизма CD28. За счет комбинации нацеленного агонизма CD28 с ТСВ мы предполагаем усилить ТСВ-опосредованные эффекторные функции, снизить порог экспрессии СЕА для эффективной ТСВ-опосредованной активации Т-клеток, обеспечить факторы выживаемости и поддержать устойчивость к супрессии Т-клеток посредством PD-1 и CTLA4.
Чтобы исследовать, могут ли нацеленные агонисты CD28 усиливать ТСВ, мы оценивали способность FAP-CD28(SA) 1+2 (P1AD9011) и FAP-CD28(SA) 1+1 (Ρ1AD4492) поддерживать ТСВ-опосредованное уничтожение клеток-мишеней. Совместное культивирование полученных из МКПК Т-клеток с коэкспрессирующими MCSP и FAP клетками MV3 в течение 5 суток в присутствии возрастающих концентраций FAP-CD28 и фиксированной ограничивающей концентрации MCSP-TCB (5 пМ) привело к повышению уничтожения клеток-мишеней MV3 в FAP-CD28 зависимым от концентрации образом (Фиг. 11А). Однако наличие ТСВ устраняет зависимость от FAP для FAP-CD28 в двухвалентном формате CD28 (P1AD9011), тогда как в одновалентном формате CD8 зависимость от FAP сохранялась (P1AD4492), что демонстрирует зависимое от концентрации повышение СЕАСАМ5-ТСВ-опосредованного уничтожения клеток-мишеней в присутствии экспрессирующих СЕА FAP-отрицательных опухолевых клеток MKN45 через 5 суток после стимуляции (Фиг. 11Б).
В альтернативном подходе мы оценивали пролиферацию Т-клеток, индуцируемую FAP-CD28 1+2 SA (P1AD9011) в присутствии или отсутствие ТСВ и в присутствии или отсутствие FAP, соответственно. Как показано на Фиг. 12А и в предыдущем примере, FAP-CD28(SA) 2+1 сильно зависит от присутствия FAP в отношении активации Т-клеток в отсутствие ТСВ. При этом в присутствии ТСВ, как показано на Фиг. 12Б, FAP-CD28(SA) 1+2 индуцирует повышение активации Т-клеток даже в отсутствие FAP.
Мы предположили, что ТСВ-индуцированный сигнал ТКР потенциально приводит к достаточной предварительной кластеризации сигнальных компонентов ТКР, делая, таким образом, поверхностное перекрестное связывание рецепторов CD28 на Т-клетках двухвалентными молекулами CD28 достаточным для того, чтобы вызвать костимуляцию. Мы сделали заключение, в случае подходов с комбинацией ТСВ одновалентность связывающей CD28 молекулы строго необходима для поддержания зависимости от опухолевой мишени нацеленной агонистической CD28-антигенсвязывающей молекулы.
Сравнение различных одновалентных форматов FAP-CD28 позволило выявить ряд различных функциональных форматов FAP-CD28 с наибольшей активностью для классического формата 1+1
Чтобы оценить влияние конкретного формата антитела FAP-CD28 на его способность усиливать ТСВ-опосредованную активацию Т-клеток, создавали разные форматы антигенсвязывающих молекул FAP-CD28 с одновалентным связыванием CD28, которые изображены на Фиг. 1 В, 1М, 1Н и 1П. В качестве референса использовали FAP-CD28 1+2 с двухвалентностью к CD28. Чтобы оценить функциональность этих форматов, Т-клетки МКПК инкубировали с экспрессирующими MCSP и FAP клетками MV3 в течение 5 суток в присутствии возрастающих концентраций форматов FAP-CD28 вместе с фиксированной ограничивающей концентрацией MCSP-TCB (5 пМ). Все форматы были способны значительно повышать пролиферацию CD8 Т-клеток (Фиг. 13А), пролиферацию CD4 Т-клеток (Фиг. 13Б) и уничтожение клеток-мишеней (Фиг. 13 В). Следует отметить, что активность молекулы С (P1AD4492) была наибольшей и сравнимой с активностью двухвалентного референсного формата CD28 1+2 SA (P1AD9011). Связывание с CD28 и FAP всех молекул показано на Фиг. 2Е и 2Ж, соответственно.
Пример 8
In vitro оценка уничтожения опухолевых клеток комбинации нацеленных на опухоль молекул CD28 и нацеленных на СЕА ТСВ
Получение Т-клеточных биспецифических антител (ТСВ) Молекулы ТСВ получали в соответствии со способами, описанными в WO 2014/131712 А1 или WO 2016/079076 А1. Получение анти-СЕА/анти-CD3 биспецифического антитела (СЕА CD3 ТСВ или СЕА ТСВ), используемого в экспериментах, описано в примере 3 в WO 2014/131712 А1. СЕА CD3 ТСВ представляет собой антитело «2+1 IgG кросс-Fab» и состоит из двух разных тяжелых цепей и двух разных легких цепей. Точечные мутации вносили в домен СНЗ («выступы во впадины»), чтобы стимулировать сборку двух разных тяжелых цепей. Обмен доменов VH и VL в CD3-связывающем Fab проводили, чтобы стимулировать правильную сборку двух разных легких цепей. 2+1 означает, что молекула имеет два антигенсвязывающих домена для СЕА и один антигенсвязывающий домен, специфический для CD3. СЕАСАМ5 CD3 ТСВ имеет такой же формат, но содержит другую связывающую СЕА молекулу и содержит точечные мутации в доменах СН и CL связывающей CD3 молекулы с целью поддержания правильного спаривания легких цепей. СЕА CD3 ТСВ содержит аминокислотные последовательности SEQ ID NO: 161, SEQ ID NO: 162, SEQ ID NO: 163 и SEQ ID NO: 164. CEACAM5 CD ТСВ содержит аминокислотные последовательности SEQ ID NO: 165, SEQ ID NO: 166, SEQ ID NO: 167 и SEQ ID NO: 168.
CEA-CD28 демонстрирует синергизм с CEACAM5-TCB в уничтожении клеток-мишеней
В альтернативном подходе мы создали биспецифическую антигенсвязывающую молекулу CEA-CD28(SA) 1+1 (молекула Μ, Р1АЕ3127) и оценили ее способность усиливать опосредованное СЕАСАМ5-ТСВ уничтожение клеток-мишеней. С этой целью экспрессирующие СЕА клетки колоректального рака MKN45 культивировали совместно с Т-клетками МКПК и CEA-CD28 (молекула Μ, Р1АЕ3127) или ненацеленным одновалентным CD28 (молекула L, P1AD8944) в присутствии или отсутствие субоптимальной концентрации СЕАСАМ5-ТСВ (10 нМ) и оценивали уничтожение клеток MKN45 в течение времени. Как показано на Фиг. 14, только комбинация СЕАСАМ5-ТСВ и CEA-CD28 приводила к уничтожению клеток-мишеней, тогда как соединения по отдельности не обеспечивали уничтожение клеток-мишеней, что указывает на эффекты синергизма. Кроме того, ненацеленный CD28 в комбинации с СЕАСАМ5-ТСВ также не индуцировал уничтожение, подчеркивая еще раз необходимость одновалентных агонистов CD28 для перекрестного связывания и, таким образом, обеспечения зависимости от опухолевой мишени.
CEA-CD28 усиливает СЕА-ТСВ и СЕАСАМ5-ТСВ и снижает порог экспрессии СЕА на раковых клетках для ТСВ для активации Т-клеток
В случае СЕА-ТСВ и СЕАСАМ5-ТСВ необходим определенный уровень экспрессии СЕА на клетках-мишенях для активации Т-клеток и уничтожения клеток-мишеней. Мы оценивали, был ли CEA-CD28 способен снижать порог экспрессии СЕА для ТСВ, чтобы индуцировать эффективное уничтожение клеток-мишеней. С этой целью Т-клетки МКПК инкубировали с возрастающими концентрациями СЕА-ТСВ (P1AD4646) или СЕАСАМ5-ТСВ (P1AD5299) и фиксированными концентрациями CEA-CD28 (молекула Μ, Р1АЕ3127) в присутствии линий клеток-мишеней с разными уровнями экспрессии СЕА:
(i) MKN45 (высокая экспрессия, прибл. 400000 СЕА-связывающих сайтов/клетка),
(ii) Lovo (средняя экспрессия, прибл. 60000 СЕА-связывающих сайтов/клетка),
(iii) НТ-29 (низкая экспрессия, прибл. 6000 СЕА-связывающих сайтов/клетка). Пролиферацию Т-клеток измеряли как показатель активации Т-клеток. как показано на Фиг. 15, CEA-CD28 мог значительно повышать активность СЕА-ТСВ и СЕАСАМ5-ТСВ. Что наиболее удивительно, хотя сами ТСВ не обеспечивали активацию Т-клеток на клетках НТ-29 с низкой экспрессией СЕА, добавление CEA-CD28 сильно повышало активность ТСВ. Мы заключили, что CEA-CD2 усиливает СЕА-ТСВ и СЕАСАМ5-ТСВ и снижает порог экспрессии СЕА на раковых клетках для ТСВ для активации Т-клеток.
Пример 9
In vitro функциональные характеристики вариантов CD28(SA) со сниженной аффинностью
Для оригинальной связывающей молекулы CD28(SA) (TGN1412) была определена аффинность KD=1 нМ. Высокоаффинные связывающие молекулы, такие как эти, подвержены риску периферических эффектов поглощения, в особенности если мишень на высоком уровне экспрессируется в периферической крови, как в случае CD28. Чтобы (i) снизить периферический эффекты поглощения и (ii) снизить риск периферической активации Т-клеток за счет связывания биспецифических нацеленных на опухоль антител к CD28 с Т-клетками вне опухоли, мы создали серию из 31 связывающей CD28 молекулы со сниженной аффинностью путем внесения точечных мутаций в CDR (смотрите пример 3). На Фиг. 4А, 4Б и 4 В показано связывание с CD28 на клетках СНО для CD28-моноспецифических одновалентных IgG из супернатантов, подтверждая, что добавление точечных мутаций создало широкий спектр связывающих молекул с различными связывающими свойствами. На основании этих данных для преобразования в биспецифический формат FAP-CD28 был отобран короткий перечень из 11 кандидатов (смотрите пример 4, таблицу 6) для дополнительного изучения характеристик. Анализ связывания подтвердил положительное связывание с FAP на клетках (Фиг. 4Е и 4Ж), а также положительное и переменное связывание выбранных вариантов с CD28 (Фиг. 4Г и 4Д).
Связывающие CD28 варианты со сниженной аффинностью являются функциональными in vitro в биспецифическом формате FAP-CD28
Чтобы оценить, были ли связывающие CD28 варианты со сниженной аффинностью функциональными и способными поддерживать ТСВ-опосредованные эффекторные функции, мы оценивали пролиферацию Т-клеток в комбинации ТСВ. С этой целью Т-клетки МКПК культивировали совместно с экспрессирующими MCSP и FAP клетками MV3 в течение 5 суток в присутствии возрастающих концентраций FAP-CD28 и фиксированной ограничивающей концентрации MCSP-TCB (5 пМ). Как изображено на Фиг. 4И, все варианты связывающих CD28 молекул были функциональными и способными повышать ТСВ-опосредованную пролиферацию Т-клеток зависимым от концентрации образом. Следует отметить, что вариант с наименьшей аффинностью 8 (Р1АЕ3131) демонстрирует приблизительно 20-кратное снижение аффинности по сравнению с родительским клоном CD28, но сохраняет приблизительно 86% его активности (Фиг. 4К). В соответствии с этими результатами все варианты могли дополнительно повышать ТСВ-опосредованное уничтожение клеток-мишеней MV3 (Фиг. 4Л). Соответствующие значения ЕС*50 приведены в таблице 8 ниже.
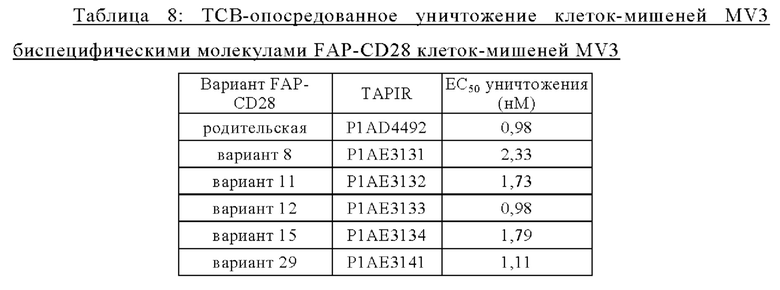
На основании этих результатов варианты 8 (наименьшая аффинность, 23 нМ), 15 (промежуточная аффинность: 7,1 нМ) и 29 (удаленные горячие точки, практически отсутствует снижение аффинности, KD=1,1 нМ) были отобраны для дополнительного изучения характеристик in vitro и исследования in vivo, оцениваемого по эффективности и улучшению биораспределения в опухоль.
Связывающие CD28 варианты со сниженной аффинностью являются функциональными in vitro в биспецифическом формате CEA-CD28
В альтернативном подходе мы преобразовали три отобранных варианта 8, 15 и 29 в нацеленный на СЕА биспецифический формат и оценили их способность повышать опосредованные СЕАСАМ5-ТСВ активацию Т-клеток и уничтожение клеток-мишеней. Связывание этих молекул с CD28 показано на Фиг. 16. Чтобы оценить функциональность, Т-клетки МКПК инкубировали с возрастающими концентрациями СЕАСАМ5-ТСВ и фиксированными концентрациями вариантов CEA-CD28 в присутствии экспрессирующих СЕА клеток-мишеней MKN45. Как изображено на Фиг. 17А, 17Б и 17В, все варианты были способны повышать ТСВ-опосредованную пролиферацию CD8 Т-клеток через 5 суток (Фиг. 17А), CD4 Т-клеток через 5 суток (Фиг. 17Б) и уничтожение клеток-мишеней через 90 ч (Фиг. 17В). Соответствующие значения ЕС50 обобщены в таблице 9 ниже.
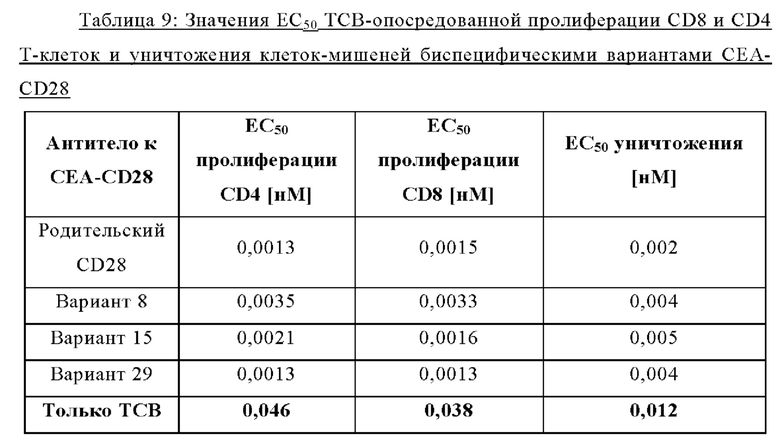
Пример 10
Создание и выработка новых анти-СЕА антител
10.1 Создание гуманизированных вариантов анти-СЕА антитела А5В7
10.1.1 Методология
Анти-СЕА антитело А5 В7 описано, например, в М. J. Banfield et al, Proteins 1997, 29(2), 161-171, а его структуру можно найти как PDB ID:1CLO в базе данных белковых структур PDB (www.rcsb.org, Н.М. Berman et al, The Protein Data Bank, Nucleic Acids Research, 2000, 28, 235-242). Эта запись включает последовательность вариабельного домена тяжелой и легкой цепи. Для идентификации подходящей человеческой акцепторной каркасной области во время гуманизации анти-СЕА связывающей молекулы А5В7, использовали классический подход путем поиска акцепторной каркасной области с высокой гомологией последовательности, прививания в эту каркасную область CDR и оценки, какие обратные мутации можно рассмотреть. Точнее, каждое аминокислотное отличие идентифицированных каркасных областей и родительского антитела оценивали в отношении влияния на целостность структуры связывающей молекулы и в случае необходимости вносили обратные мутации в направлении родительской последовательности. Структурная оценка основывалась на моделях гомологии Fv-области как родительского антитела, так и его гуманизированных версий, созданных с помощью внутрилабораторного инструмента моделирования гомологии структуры антител, реализуемого с применением Biovia Discovery Studio Environment версии 4.5.
10.1.2 Выбор и адаптация акцепторной каркасной области
Акцепторная каркасная область была выбрана, как описано в таблице 10
ниже:
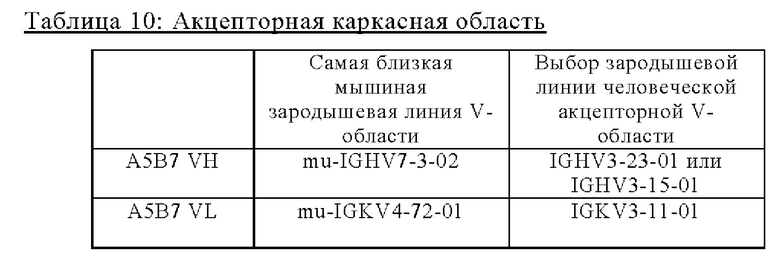
Каркасные области после CDR3 были адаптированы из IGJH6 зародышевой линии J-элементов человека для тяжелой цепи и последовательности, сходной с IGKJ2 J-элемента каппа, для легкой цепи.
На основании структурных соображений в позиции 93 и 94 тяжелой цепи были внесены обратные мутации с человеческой акцепторной каркасной области на аминокислоту в родительской связывающей молекуле.
10.1.3 Области VH и VL полученных в результате гуманизированных антител к СЕА
Полученные в результате домены VH гуманизированных антител к СЕА можно найти в таблице 11 ниже, а полученные в результате домены VL гуманизированных антител к СЕА перечислены в таблице 12 ниже.

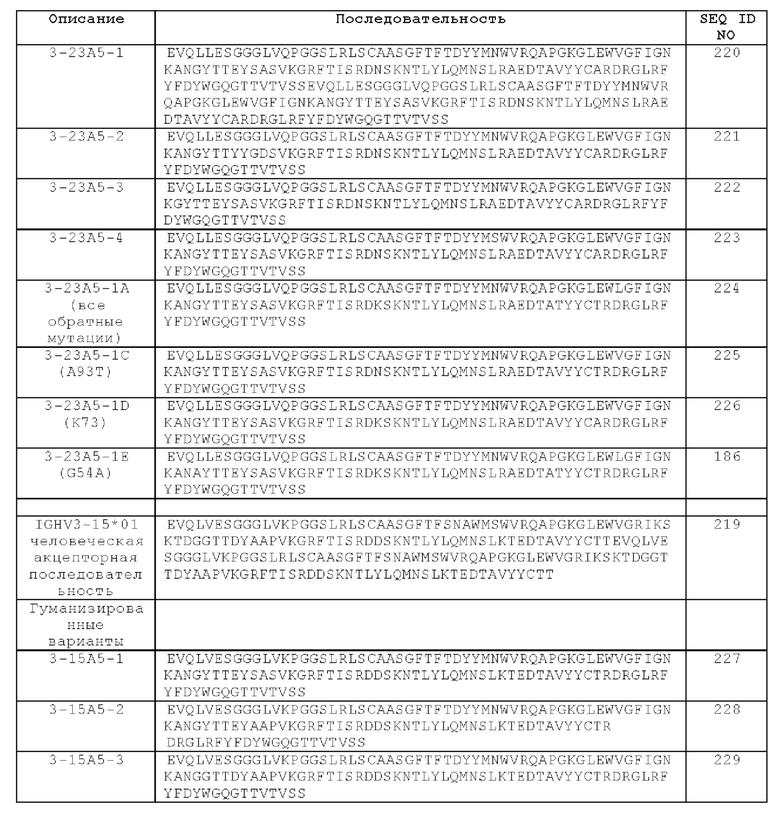
Для тяжелой цепи исходный вариант 3-23А5-1 был найден подходящим в анализе связывания (но демонстрировал слегка меньшее связывание, чем родительское антитело) и был выбран в качестве отправной точки для дополнительных модификаций. Варианты на основании IGHV3-15 демонстрировали меньшую активность связывания по сравнению с гуманизированным вариантом 3-23А5-1.
Чтобы восстановить полную активность связывания родительского химерного антитела, создали варианты 3-23А5-1А, 3-23А5-1С и 3-23A5-1D. Для варианта 3-23А5-1 также проводили исследование, можно ли адаптировать длину CDR-H2 к человеческой каркасной последовательности, но эта конструкция полностью утратила активность связывания. Поскольку в CDR-H2 (Asn53-Gly54) присутствовала возможная горячая точка дезамидирования, мы заменили этот мотив на Asn53-Ala54. В другой возможной горячей точке Asn73-Ser74 провели обратную мутацию на Lys73-Ser74. Таким образом был создан вариант 3-23А5-1Е.
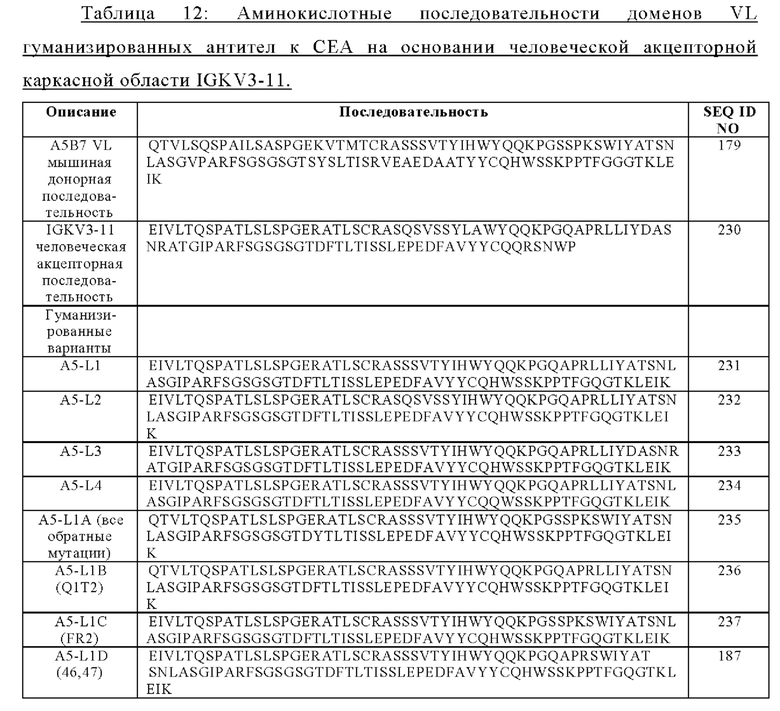
Легкую цепь гуманизировали на основании человеческой акцепторной каркасной области IGKV3-11. В серии от A5-L1 до A5-L4 было выявлено, что вариант A5-L1 демонстрирует хорошую активность связывания (но слегка меньшую, чем родительское антитело). Частичная гуманизация CDR-L1 (вариант A5-L2; позиции 30 и 31 по Kabat) полностью устраняла связывание. Аналогично, гуманизация CDR-H2 (вариант A5-L3; позиции от 50 до 56 по Kabat) также полностью устраняла связывание. Позиция 90 (вариант A5-L4) демонстрирует существенный вклад в свойства связывания. Гистидин в этой позиции важен для связывания. Таким образом, вариант A5-L1 был выбран для дополнительной модификации.
Серия от A5-L1A до A5-L1D решила вопрос, какие обратные мутации необходимы для восстановления полного связывающего потенциала родительского химерного антитела. Вариант A5-L1A показал, что обратные мутации в позициях 1, 2 по Kabat, всей каркасной области 2 и позиции 71 по Kabat не обеспечивают какую-либо дополнительную активность связывания. Варианты A5-L1B и A5-L1C решили вопросы, связанные с подгруппами этих позиций и подтвердили, что они не меняют свойств связывания. Вариант А5-L1D с обратными мутациями в позициях 46 и 47 по Kabat продемонстрировал наилучшую активность связывания.
10.1.4 Отбор гуманизированных антител А5В7
На основании новых вариантов гуманизации VH и VL новые антитела к СЕА экспрессировали в виде антител huIgG1 с сайленсингом эффектора Fc (P329G; L234, L235A) для устранения связывания с Fcγ-рецепторами в соответствии со способом, описанным в WO 2012/130831 А1, и исследовали их связывание с СЕА, экспрессируемым на клетках MKN45, и сравнивали с соответствующим родительским мышиным антителом А5В7.
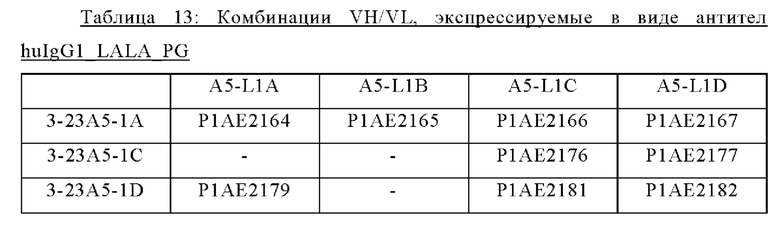
MKN45 (DSMZ АСС 409) представляет собой линию клеток аденокарциномы желудка, экспрессирующую СЕА. Эти клетки культивировали в расширенной среде RPMI+2% ФТС+1% Glutamax. Проверяли жизнеспособность клеток MKN-45 и ресуспендировали клетки и доводили до плотности 1 млн клеток/мл. 100 мкл этой клеточной суспензии (содержащие 0,1 млн клеток) высевали в 96-луночный круглодонный планшет. Планшет центрифугировали в течение 4 мин при 400xg и удаляли супернатант. Потом в клетки добавляли 40 мкл разведенных антител или буфера FACS и инкубировали в течение 30 мин при 4°С. После инкубации клетки дважды промывали 150 мкл буфера FACS на лунку. Потом в клетки добавляли 20 мкл разведенного вторичного РЕ античеловеческого Fc-специфического вторичного антитела (109-116-170, Jackson ImmunoResearch). Клетки инкубировали еще в течение 30 мин при 4°С. Для удаления несвязанного антитела клетки снова дважды промывали 150 мкл буфера FACS на лунку. Для фиксации клеток в лунки добавляли 100 мкл буфера FACS, содержащего 1% ПФА. Перед измерением клетки ресуспендировали в 150 мкл буфера FACS. Флуоресценцию измеряли, используя проточный цитометр BD.
На Фиг. 18 приведены кривые связывания гуманизированных вариантов А5В7. Все исследуемые связывающие молекулы были способны связываться с клетками MKN45, но связывающая способность была слегка снижена по сравнению с родительским антителом А5В7. Клон Р1АЕ2167 имел наилучшее связывание из всех исследованных вариантов и был отобран для дополнительной разработки.
10.1.5 Определение аффинности Fab-фрагментов гуманизированных вариантов мышиного антитела к СЕА А5В7 с СЕА человека с помощью поверхностного плазмонного резонанса (BIACORE)
Аффинность Fab-фрагментов гуманизированных вариантов мышиного антитела к СЕА А5В7 с СЕА человека оценивали методом поверхностного плазмонного резонанса, используя инструмент BIACORE Т200. На чипе СМ5 иммобилизовали СЕА человека (hu N(A2-B2)A-avi-His В) в концентрации 40 нМ посредством стандартной реакции сопряжения аминов на проточной ячейке 2 в течение 30 с до около 100 ЕО. После этого в качестве аналитов вводили Fab-фрагменты гуманизированных вариантов мышиного антитела к СЕА А5В7 в 3-кратных разведениях в диапазоне 500-0,656 нМ в течение времени контакта 120 с, со временем диссоциации 250 или 1000 с при скорости потока 30 мкл/мин. Восстановление на уровне СЕА человека (hu N(A2-B2)A-avi-His В) обеспечивали 2 импульсами 10 мМ глицина/HCl, рН 2,0, в течение 60 с. Данные проходили двойной сравнение относительно иммобилизованной проточной ячейки 1 и нулевой концентрации аналита. Сенсограммы аналитов аппроксимировали простой 1:1 моделью взаимодействия Лэнгмюра. Константы аффинности [KD] для СЕА человека (домен А2) обобщены в таблице 14 ниже.
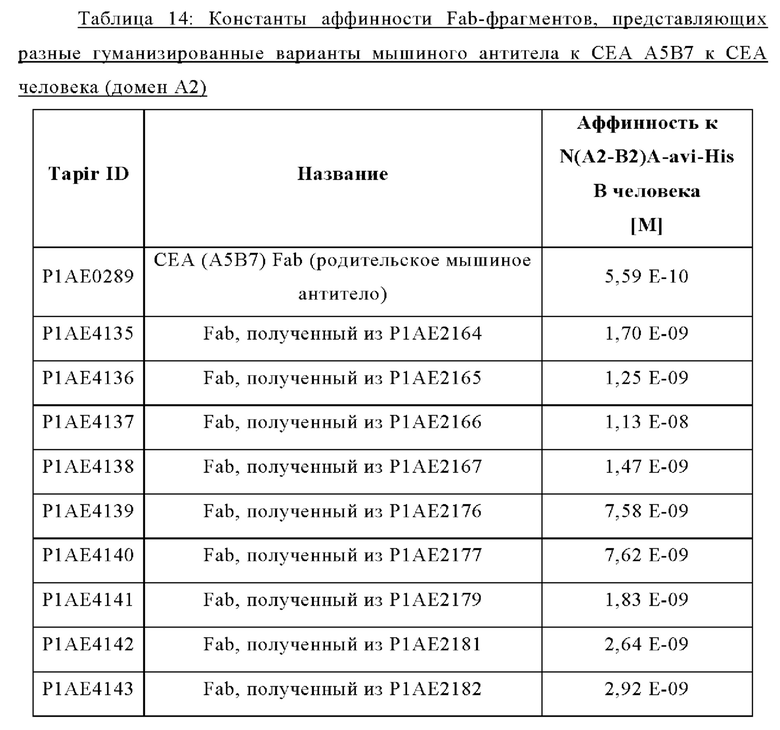
Гуманизированные варианты мышиного антитела к СЕА А5В7 имеют меньшую Аффинность, чем родительское мышиное антитело. Fab-фрагмент Р1АЕ4138, полученный из Р1АЕ2167 (тяжелая цепь с вариантом VH 3-23А5-1А и легкая цепи С-каппа с вариантом VL A5-L1D) был выбран как конечный гуманизированный вариант. Кроме того, мутация с глицина на аланин в позиции 54 по Kabat (G54A) была внесена в домен VH для удаления сайта дезамидирования, что дало вариант VL 3-23А5-1Е. Конечное гуманизированное антитело (тяжелая цепь с вариантом VH 3-23А5-1Е и легкая цепи С-каппа с вариантом VL A5-L1D) было названо A5H1EL1D или huA5B7.
10.2 Создание полученных из A5H1EL1D анти-СЕА антител с созревшей аффинностью
10.2.1 Получение, очистка и изучение характеристик антигенов для подхода фагового дисплея
Мышиное антитело А5В7 и его гуманизированное производное A5H1EL1D связываются с доменом А2 СЕАСАМ5 (СЕА) с аффинностью около 0,8 и около 2,5 нМ, соответственно. Для создания вариантов A5H1EL1D с созревшей аффинностью методом фагового дисплея создали 3 разных рекомбинантных растворимых антигена. Каждый белок содержал С-концевой avi-тег для сайт-специфического биотинилирования и his-тег для очистки: Первый белок состоял из внеклеточной части СЕАСАМ1, состоящей из 4 Ig-подобных доменов Ν, Α1, В, А2 (NABA-avi-His, SEQ ID NO: 238, таблица 15). Второй белок представлял собой химерный белок, состоящий из 2 Ig-доменов СЕАСАМ5 и 2 СЕАСАМ1. На основании последовательности четырех доменов СЕАСАМ1, ДНК, кодирующую второй и третий домены СЕАСАМ1 (домены А1 и В), замещали ДНК, кодирующей домены А2 и В2 СЕАСАМ5 (N(A2B2)A-avi-His, SEQ ID NO:239, таблица 15). Третий белок представлял собой химерный белок, состоящий из 1 Ig-домена СЕАСАМ5 и 3 СЕАСАМ1. На основании последовательности четырех доменов СЕАСАМ1, ДНК, кодирующую третий домен CEACAM1 (домены В), замещали ДНК, кодирующей домен В2 СЕАСАМ5 (NA(B2)A-avi-His, SEQ ID N0:240, таблица 15). Схематическое описание трех конструкций приведено на Фиг. 19А, 19Б и 19В.


Соответствующие плазмиды временно трансфицировали в клетки HEK 293, стабильно экспрессирующие полученный из EBV белок EBNA (HEK EBNA). Одновременно котрансфицируемая плазмида, кодирующая биотинлигазу BirA, делала возможным avi-тег-специфическое биотинилирование in vivo. Белки очищали из профильтрованных клеточных культуральных супернатантов согласно стандартным протоколам, используя аффинную хроматографию на иммобилизованных ионах металла (IMAC) с последующей гель-фильтрацией. Фракции мономерных белков объединяли, концентрировали (при необходимости), замораживали и хранили при -80°С. Часть образцов предоставляли для последующего белкового анализа и аналитического определения, например, с помощью ДСН-ПААГ, эксклюзионной хроматографии (ЭХ) или масс-спектрометрии.
10.2.2 Отбор полученных из A5H1EL1D антител с созревшей аффинностью
Гуманизация антитела А5В7 привела к около 3-4-кратному снижению аффинности к СЕА по данным измерения ППР. Хотя аффинность в отношении А5В7 составляла около 0,8 нМ, для A5H1EL1D была измерена аффинность около 2,5 нМ. Эксперименты FACS с использование линий клеток с разными уровнями экспрессии СЕА подтвердили это наблюдение. Чтобы улучшить аффинность гуманизированного клона A5H1EL1D, создали 3 разные библиотеки созревания аффинности, которые использовали для отбора клонов с улучшенной аффинностью методом фагового дисплея.
10.2.2.1 Создание библиотек A5H1EL1D созревания аффинности Создание полученных из A5H1EL1D антител с созревшей аффинностью проводили методом фагового дисплея, используя стандартные протоколы (Silacci et al, 2005). На первом этапе последовательности ДНК, кодирующие VH и VL гуманизированного родительского клона A5H1EL1D (аминокислотные последовательности SEQ ID NO: 186 и 187) клонировали в фагмиду, которую затем использовали в качестве матрицы для рандомизации. На следующем этапе создавали три библиотеки для отбора подходящих клонов методом фагового дисплея. Библиотеки созревания 1 и 2 рандомизировали в CDR1 и CDR2 тяжелой цепи или в CDR1 и CDR2 легкой цепи. Третью библиотеку созревания рандомизировали в областях CDR3 как тяжелой, так и легкой цепи. Рандомизированные позиции в соответствующих областях CDR показаны на Фиг. 20А и 20Б. Для создания библиотеки созревания 1, рандомизированной в CDR1 и 2 тяжелой цепи, проводили сборку двух фрагментов методом ПЦР со «сплайсингом путем перекрытия удлинений» (SOE) и клонировали в фаговый вектор (Фиг. 21А). Для создания фрагментов библиотеки использовали следующие комбинации праймеров: фрагмент 1 (LMB3 (SEQ ID NO: 243, таблица 16) и A5H1EL1D_H1_rev_TN (SEQ ID NO: 241, таблица 16) и фрагмент 2 (A5H1EL1D_Н2_прям_TN (SEQ ID NO: 242, таблица 7) и HCDR3-обратн-констант (SEQ ID NO: 244, таблица 16).
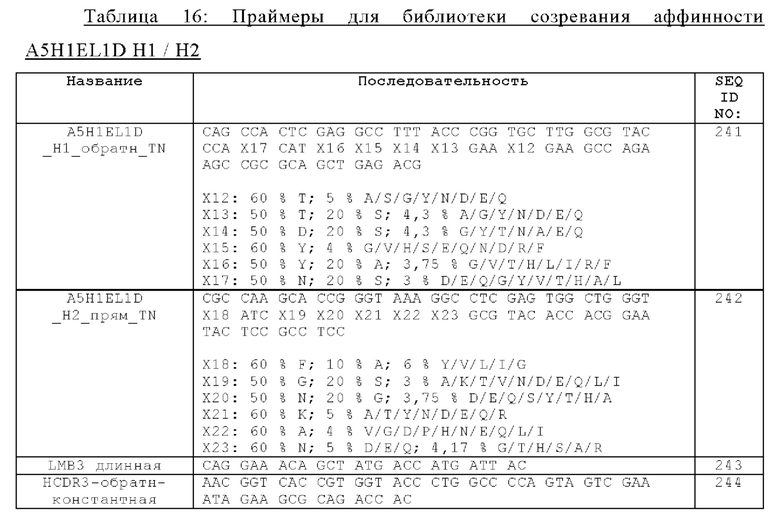
Для создания библиотеки созревания 2, рандомизированной в CDR1 и 2 легкой цепи, проводили сборку двух фрагментов методом ПЦР со «сплайсингом путем перекрытия удлинений» (SOE) и клонировали в фаговый вектор (Фиг. 21Б). Для создания фрагментов библиотеки использовали следующие комбинации праймеров: фрагмент 1 (LMB3 (SEQ ID NO: 243, таблица 17) и A5H1EL1D_L1_rev_TN (SEQ ID NO: 245, таблица 17) и фрагмент 2 (A5H1EL1D_L2_прям_TN (SEQ ID NO: 246, таблица 17) и HCDR3-обратн-констант (SEQ ID NO: 244, таблица 17).
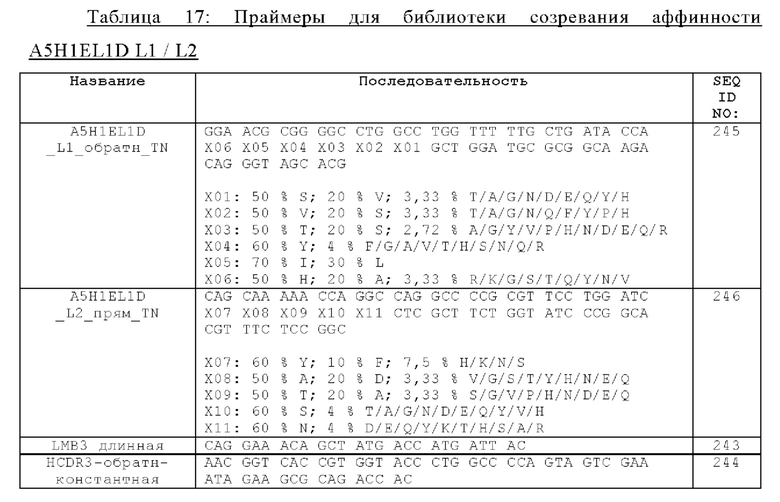
Для создания библиотеки созревания 3, рандомизированной в CDR3 легкой и тяжелой цепей, проводили сборку двух фрагментов методом ПЦР со «сплайсингом путем перекрытия удлинений» (SOE) и клонировали в фаговый вектор (Фиг. 21В). Для создания фрагментов библиотеки использовали следующие комбинации праймеров: фрагмент 1 (LMB3 (SEQ ID NO: 243, таблица 18) и LCDR3-обратн-констант (SEQ ID NO: 249, таблица 18) и фрагмент 2 (A5H1EL1D_L3_прям_TN (SEQ ID NO: 247, таблица 18) и A5H1EL1D_H3_обратн_TN (SEQ ID NO: 248, таблица 18).


Для сборки фрагментов каждой библиотеки использовали эквимолярные количества каждого фрагмента и амплифицировали с соответствующими внешними праймерами. Для сборки фрагментов третьей библиотеки, рандомизированной в HCDR3 и LCDR3, праймер LMB3 (SEQ ID NO:243, таблица 18) использовали в комбинации с праймером « амплификация HCDR3» (SEQ ID NO: 250, таблица 18). Этот праймер использовали для продления С-конца VH последовательностью, содержащей сайт ΚpnΙ. После сборки достаточного количества полноразмерных фрагментов для всех библиотек их расщепляли NcoI/KpnI наряду с идентично обрабатываемым акцепторным фагмидным вектором. 3-Кратный молярный избыток библиотечной вставки лигировали 20 μг фагмидного вектора. Очищенное лигирование использовали для 20 трансформаций, что привело к получению от около 0,7 x 109 до 2 x 109 трансформантов. Фагмидные частицы, отображающие библиотеки созревания аффинности A5H1EL1D, изымали и проводили очистку ПЭГ/NaCl для применения в отборе.
10.2.2.2 Отбор полученных из A5H1EL1D клонов с созревшей аффинностью
Для отбора клонов с созревшей аффинностью проводили отбор методом фагового дисплея со всем 3 библиотеками, используя рекомбинантные растворимые антигены. Раунды пэннинга проводили в растворе в соответствии со следующей схемой: 1. Предварительная очистка неспецифических фагмидных частиц путем инкубации с 200 нМ биотинилированного NA(B2)A-avi-His τ NABA-avi-his в течение 0,5 ч, 2. Захват биотинилированного NA(B2)A-avi-His, NABA-avi-his и связанных фагмидных частиц путем добавления 5,4 x 107 покрытых стрептавидином магнитных гранул в течение 10 мин, 3. Выделение несвязанных фагмидных частиц из супернатанта для дополнительного отбора, 4. связывание фагмидных частиц с 20 нМ биотинилированного N(A2B2)A-avi-His в течение 0,5 ч в общем объеме 1 мл, 5. захват биотинилированного белка N(A2B2)A-avi-His и специфически связанных фаговых частиц путем добавления 5,4 x 107 покрытых стрептавидином магнитных гранул в течение 10 мин, 6. промывка гранул с использованием 5 x 1 мл ФСБ/Твин 20 и 5 x 1 мл ФСБ, 7. элюирование фаговых частиц путем добавления 1 мл 100 мМ ТЭА в течение 10 мин и нейтрализация путем добавления 500 μл 1 Μ Трис/HCl, рН 7,4, 8. инфицирование экспоненциально растущими бактериями Е. coli TG1, 9. инфицирование хелперным фагом VCSM13 и 10. последующее осаждение ПЭГ/NaCl фагмидных частиц для применения в последующих раундах отбора. Отбор проводили в течение 3 раундов, используя снижающиеся концентрации антигена (20 x 10-9 Μ, 10 x 10-9 Μ и 2 x 10-9 Μ). На 3 раунде стрептавидиновые гранулы промывали 20 x 1 мл ФСБ/Твин 20 и 5 x 1 мл ФСБ.
Специфические связывающие молекулы идентифицировали методом ELISA следующим образом: 100 μл 10 нМ биотинилированного белка N(A2B2)A-avi-His или 40 нМ биотинилированного белка NA(B2)A-avi-His на лунку наносили на нейтравидиновые планшеты. Добавляли Fab-содержащие бактериальные супернатанты и проводили обнаружение Fab посредством Flag-тегов, используя вторичное анти-Flag/HRP антитело. Клоны, которые были ELISA-положительными на рекомбинантном белке N(A2B2)A-avi-His, но не на белке NA(B2)A-avi-His, дополнительно исследовали методом ППР.
10.2.2.3 Идентификация полученных из A5H1EL1D вариантов с созревшей аффинностью методом ППР
Чтобы получить дополнительные характеристики ELISA-положительных клонов, измеряли скорость диссоциации методом поверхностного плазмонного резонанса, используя прибор Proteon XPR36, и сравнивали результаты с родительским положительным клоном A5H1EL1D.
Для этого эксперимента около 2000, 1000 и 500 ЕО биотинилированного N(A2B2)A-avi-His иммобилизовали на 3 каналах, используя покрытый стрептавидином чип NLC в вертикальной ориентации. В качестве контроля неспецифического связывания 2000 ЕО биотинилированного белка NA(B2)A-avi-His иммобилизовали на канале 4. Для анализа скорости диссоциации идентифицированных ELISA-положительных клонов направление введения меняли на горизонтальную ориентацию. Перед введением каждый Fab-содержащий бактериальный супернатант фильтровали и в 3 раза разводили ФСБ. Время ассоциации составляло 100 с при 100 μл/мин, а время диссоциации составляло 600 или 1200 с. Бактериальные супернатанты без Fab-фрагмента использовали как референс.Восстановление проводили 10 мМ глицина, рН 1,5, в течение 35 с при 50 μл/мин (вертикальная ориентация).
Значения скорости диссоциации (koff) рассчитывали, используя простую один-к-одному модель связывания Лэнгмюра в программном обеспечении ProteOn Manager v3.1, путем одновременной аппроксимации сенсограмм. Идентифицировали клоны, экспрессирующие Fab с наименьшей скоростью диссоциации, и считали прошедшими отбор. Прошедшие отбор клоны повторно оценивали в дополнительных экспериментах ППР в тех же условиях. На это раз во время каждого введения 4 клона с созревшей аффинностью сравнивали напрямую параллельно с родительским клоном A5H1EL1D. Бактериальные супернатанты без Fab-фрагмента использовали как референс.Отбирали клоны, которые демонстрировали меньшую скорость диссоциации, чем A5H1EL1D, на N(A2B2)A-avi-His и не связывались NA(B2)A-avi-His, и секвенировали вариабельные домены соответствующих фагмид. Измеренные скорости диссоциации наилучших клонов приведены в таблице 19, а последовательности соответствующих вариабельных доменов приведены в таблице 20.
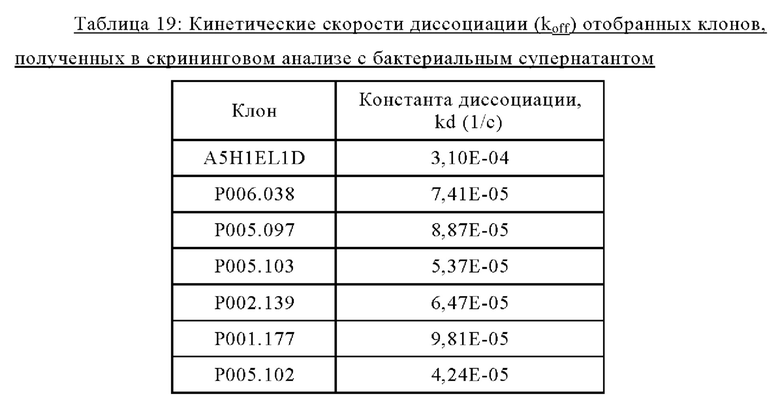
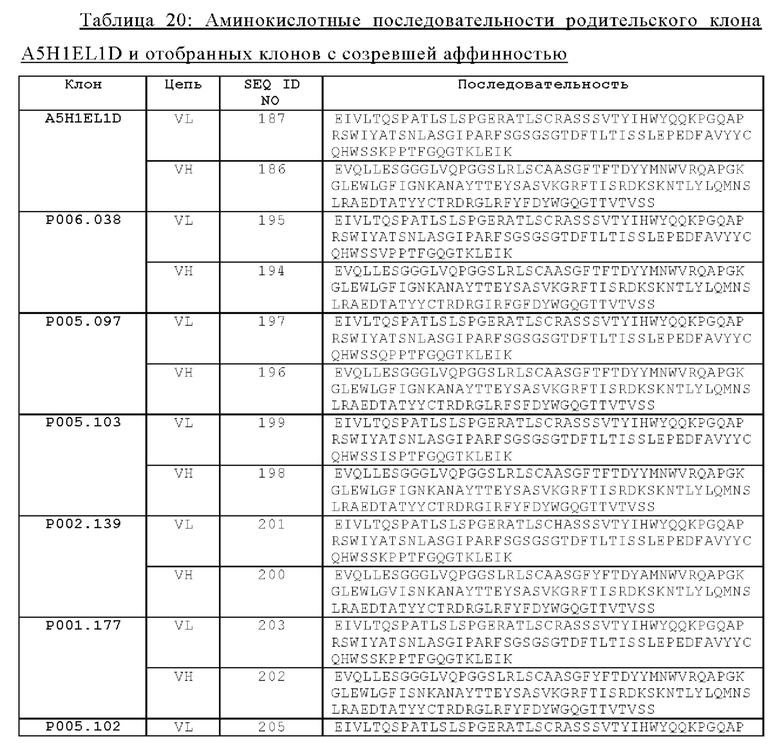
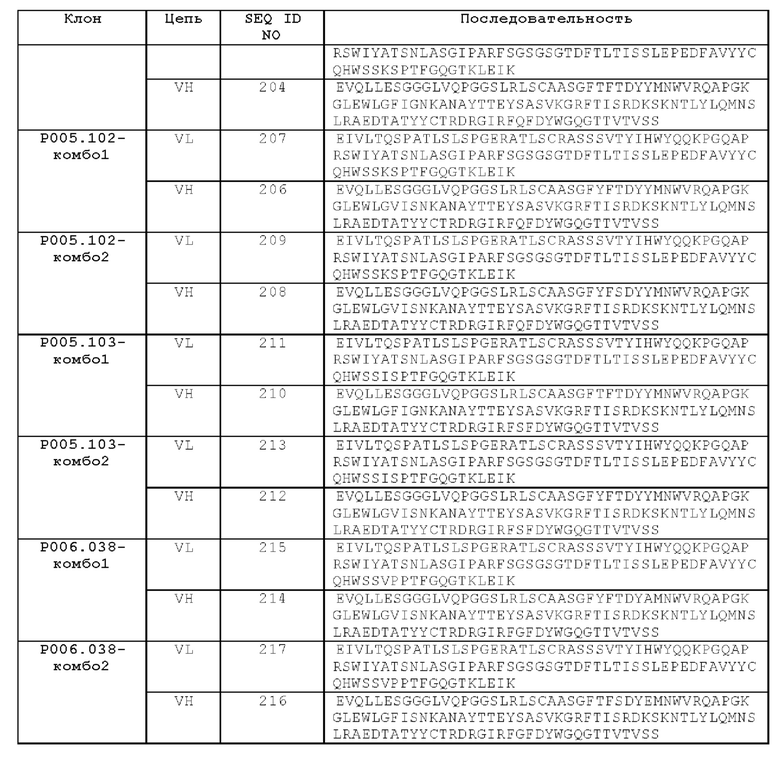
10.2.2.4 Очистка Fab клонов A5H1EL1D с созревшей аффинностью
Чтобы получить дополнительные характеристики клонов с созревшей аффинностью, очищали соответствующие Fab-фрагменты для точного анализа кинетических параметров. Для каждого клона 500 мл культуры инокулировали бактериями, несущими соответствующую фагмиду, и индуцировали 1 мМ IPTG при оптической плотности, измеряемой на 600 нм (ОП600), 0,9. После этого культуры инкубировали при 25°С в течение ночи и собирали путем центрифугирования. После инкубации ресуспендированного осадка в течение 20 мин в 25 мл буфера ФСБ (30 мМ Трис-HCl, рН8, 1 мМ ЭДТА, 20% сахарозы), бактерии снова центрифугировали и собирали супернатант. Этот этап инкубации повторяли один раз с 25 мл 5 мМ раствора MgSO4. Супернатанты обоих этапов инкубации объединяли, фильтровали и загружали на колонку IMAC (His gravitrap, GE Healthcare). После этого колонку промывали 40 мл промывочного буфера (500 мМ NaCl, 20 мМ имидазола, 20 мМ NaH2PO4, рН 7,4). После элюирования (500 мМ NaCl, 500 мМ имидазола, 20 мМ NaH2PO4, рН 7,4) элюат повторно забуферивали, используя колонки PD10 (GE Healthcare). Выход очищенного белка находился в диапазоне от 300 до 500 μг/л.
10.2.2.5 ППР-анализ очищенных Fab-фрагментов A5H1EL1D с созревшей аффинностью
Аффинность (KD) очищенных Fab-фрагментов измеряли методом поверхностного плазмонного резонанса, используя прибор Proteon XPR36, используя ту же схему, которая была описана ранее.
Около 2000, 1000, 500 и 250 ЕО биотинилированного N(A2B2)A-avi-His иммобилизовали на 4 каналах покрытого стрептавидином чипа NLC в вертикальной ориентации. В качестве контроля неспецифического связывания 2000 ЕО биотинилированного белка NA(B2)A-avi-His иммобилизовали на канале 5. Для определения аффинности (KD) очищенных клонов направление введения меняли на горизонтальную ориентацию. Двукратные серийные разведения очищенных Fab-фрагментов (диапазон переменных концентраций от 100 до 3 нМ) вводили одновременно при 100 μл/мин вдоль отдельных каналов 1-5 со временем ассоциации 100 с и временем диссоциации 1200 с. Буфер (ФСБТ) вводили вдоль шестого канала для обеспечения «параллельной» холостой пробы для сравнения. Восстановление проводили 10 мМ глицина, рН 1,5, в течение 35 с при 50μл/мин (вертикальная ориентация).
Константы скорости ассоциации (kon) и константы скорости диссоциации (koff) рассчитывали, используя простую один-к-одному модель связывания Лэнгмюра в программном обеспечении ProteOn Manager v3.1, путем одновременной аппроксимации сенсограмм ассоциации и диссоциации. Равновесную константу диссоциации (KD) рассчитывали как соотношение koff/kon. Кинетические и термодинамические данные приведены в таблице 21.
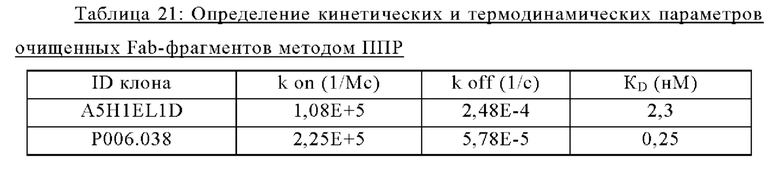
10.2.2.6 Комбинация позиций CDR клонов с созревшей аффинностью
В попытке дополнительно повысить аффинность к СЕА позиции CDR нескольких ранее идентифицированных связывающих молекул с созревшей аффинностью комбинировали друг с другом. Это включает не только конкретные позиции в CDR, но также комбинации CDR из разных связывающих молекул. Выравнивание всех клонов, полученных из фагового дисплея и комбинаторных клонов, показано на Фиг. 22А и 22Б. CDR всех тяжелых цепей и легких цепей перечислены в таблицах 22 и 23, соответственно. Домены VH и VL обобщены в таблице 20.
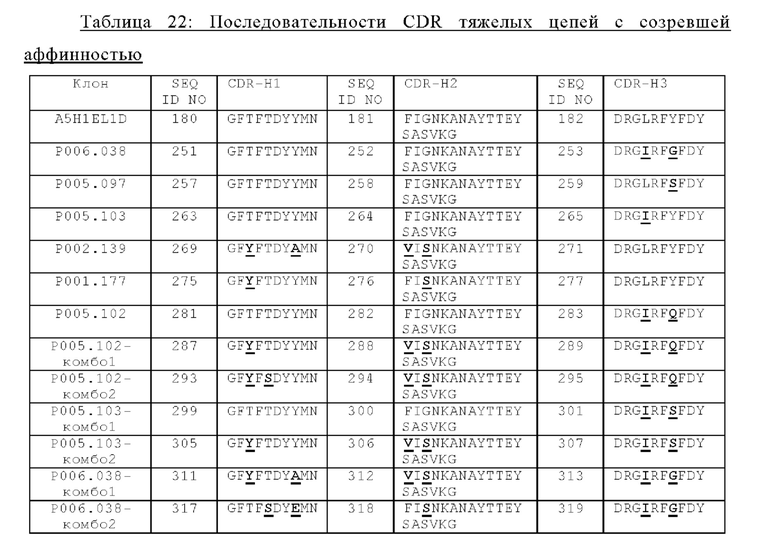
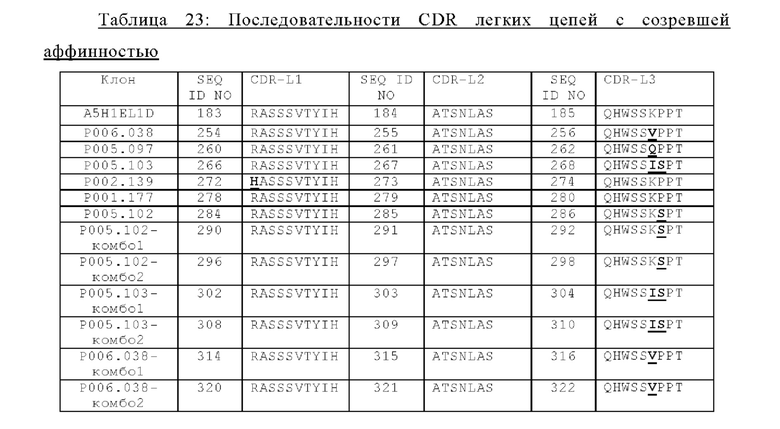
10.3 Создание и изучение характеристик полученных из A5H1EL1D биспецифических антител с созревшей аффинностью
10.3.1 Клонирование, выработка и очистка биспецифических антител
Вариабельные домены всех клонов синтезировали и клонировали в плазмиды, кодирующие биспецифическую молекулу 1+1 IgG, на основании мутаций типа «выступ во впадину» и технологии crossmab в комбинации с мутациями PG-LALA. Второй связывающий фрагмент был специфичен в отношении CD28. Схематическое описание конечных молекул изображено на Фиг. 1Л. Полученные в результате молекулы состояли из последовательностей, перечисленных в таблице 24: Все комбинации цепей, экспрессирующие последовательности VL и VH связывающих молекул с созревшей аффинностью (SEQ ID NO: 323-348), комбинировали с двумя цепями, специфическими к CD28 (SEQ ID NO: 349 и 350).
Полученные в результате конструкции были изготовлены Evitria с использованием их собственной векторной системы с помощью стандартных (не на основе ПЦР) методик клонирования и использованием адаптированных к суспензии клеток СНО K1 (изначально полученных от АТСС и адаптированных для бессывороточного роста в суспензионной культуре в Evitria). Для выработки Evitria использовали их собственные не содержащую животных компонентов и не содержащую сыворотку среды (eviGrow и eviMake2) и их собственный трансфекционный реагент (eviFect). Белки очищали из профильтрованных клеточных культуральных супернатантов согласно стандартным протоколам. Вкратце, Fc-содержащие белки очищали из клеточных культуральных супернатантов с помощью аффинной хроматографии с протеином А. Элюирование проводили при рН 3,0 с последующей немедленной нейтрализацией образца. Белок концентрировали и отделяли агрегированный белок от мономерного белка с помощью эксклюзионной хроматографии в 20 мМ гистидине, 140 мМ хлориде натрия, рН 6,0.
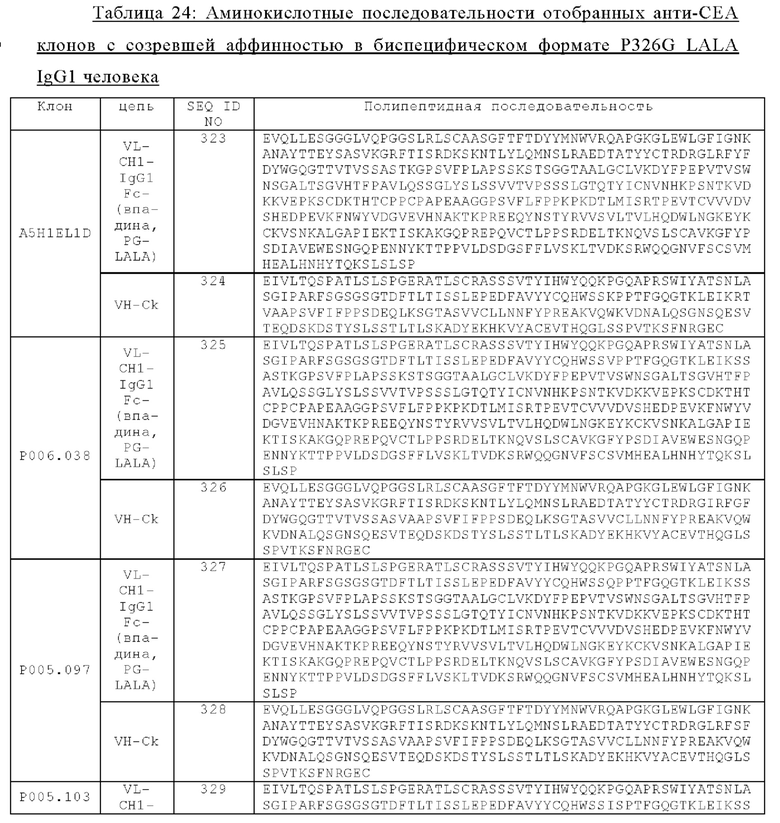
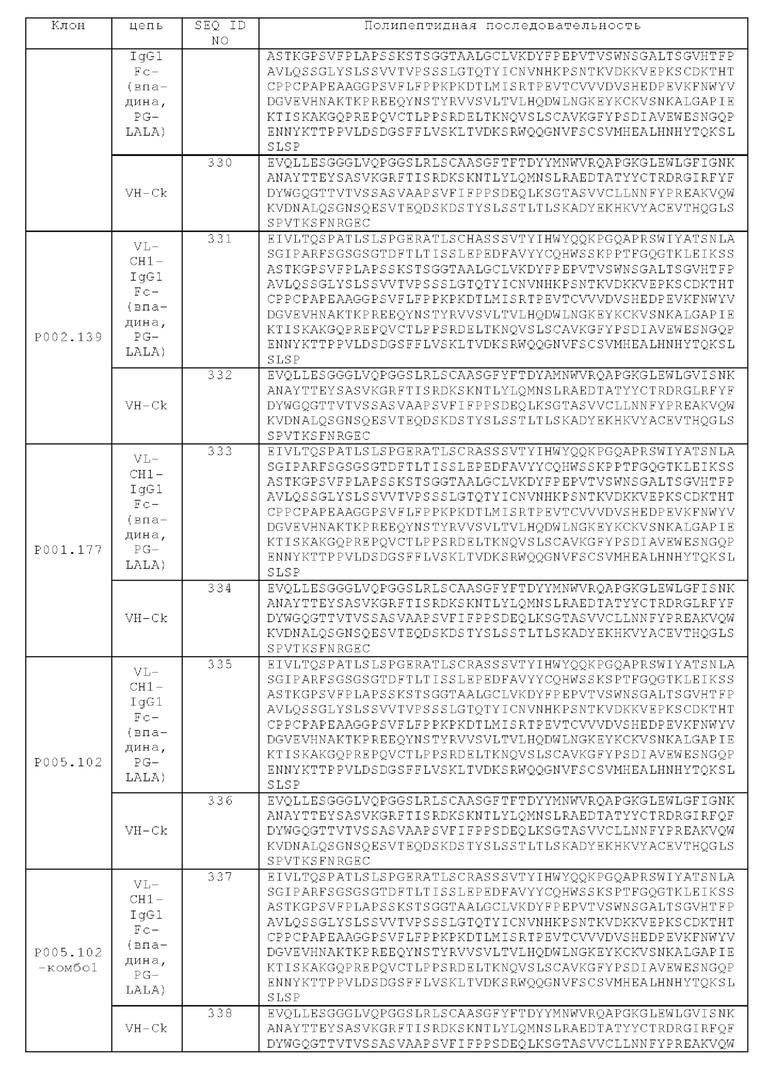
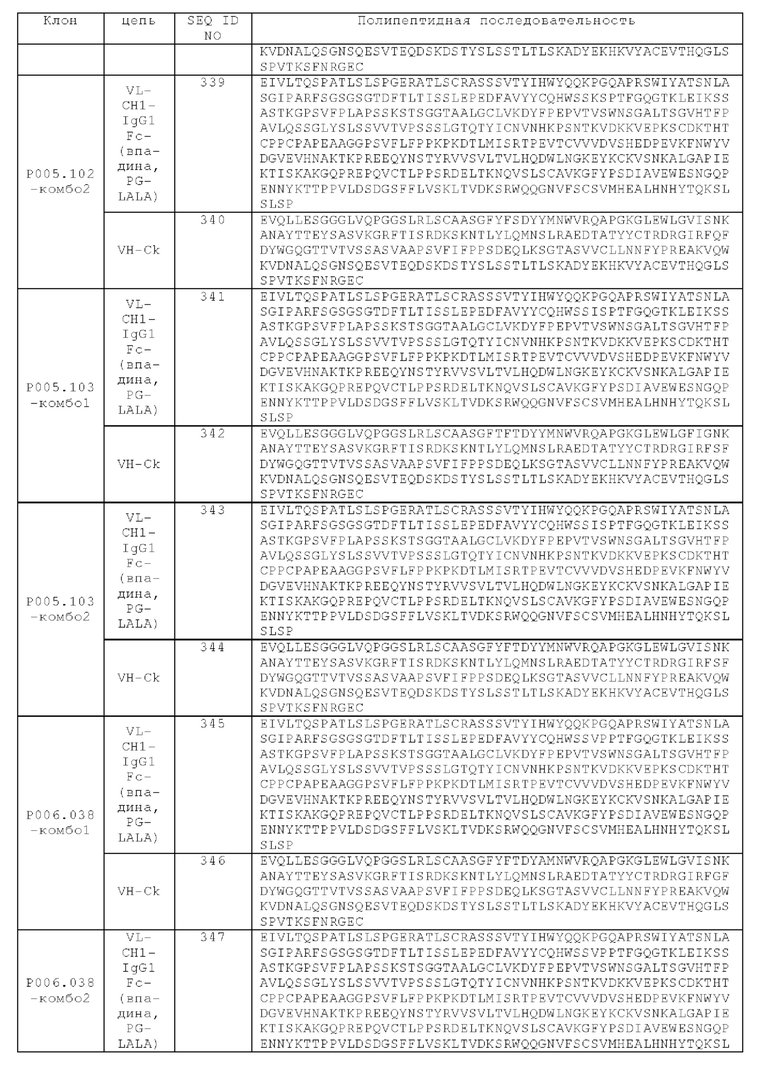
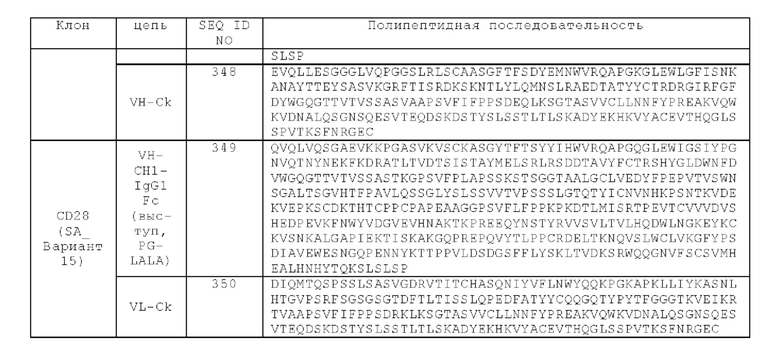
10.3.2 Определение аффинности отобранныз антител методом ППР
Аффинность (KD) родительского антитела A5H1EL1D, а также его производных с созревшей аффинностью измеряли методом ППР, используя инструмент ProteOn XPR36 (Biorad) при 25°С.
Около 2000, 1000, 500 и 250 ЕО биотинилированного N(A2B2)A-avi-His иммобилизовали на 4 каналах покрытого стрептавидином чипа NLC в вертикальной ориентации. В качестве контроля неспецифического связывания 2000 ЕО биотинилированного белка NA(B2)A-avi-His иммобилизовали на канале 5. Для определения аффинности (KD) очищенных биспецифических конструкций направление введения меняли на горизонтальную ориентацию. Двукратные серийные разведения очищенных биспецифических IgG (диапазон переменных концентраций от 25 до 1,56 нМ) вводили одновременно при 100 мкл/мин вдоль отдельных каналов 1-5 со временем ассоциации 180 с и временем диссоциации 1200 с. Буфер (ФСБТ) вводили вдоль шестого канала для обеспечения «параллельной» холостой пробы для сравнения. Восстановление проводили 10 мМ глицина, рН 1,5, в течение 20 с при 50 мкл/мин (вертикальная ориентация).
Константы скорости ассоциации (kon) и константы скорости диссоциации (koff) рассчитывали, используя простую один-к-одному модель связывания Лэнгмюра в программном обеспечении ProteOn Manager v3.1, путем одновременной аппроксимации сенсограмм ассоциации и диссоциации. Равновесную константу диссоциации (KD) рассчитывали как соотношение koff/kon. Все кинетические и термодинамические данные приведены в таблице 25.
Большую аффинность (меньшие значения KD) наблюдали для клонов с созревшей аффинностью, которые были идентифицированы при отборе методом фагового дисплея. Кроме того, исследовали комбинации с обменом CDR и позиций CDR. Хотя некоторые комбинации (например, клоны «Р005.103-комбо2» и «Р005.102-комбо1») демонстрировали очень медленные скорости диссоциации и, следовательно, очень высокую аффинность, аффинность была существенно снижена в 2 комбинаторных клонах (клоны «Р006.038-комбо1» и «Р006.038-комбо2»).
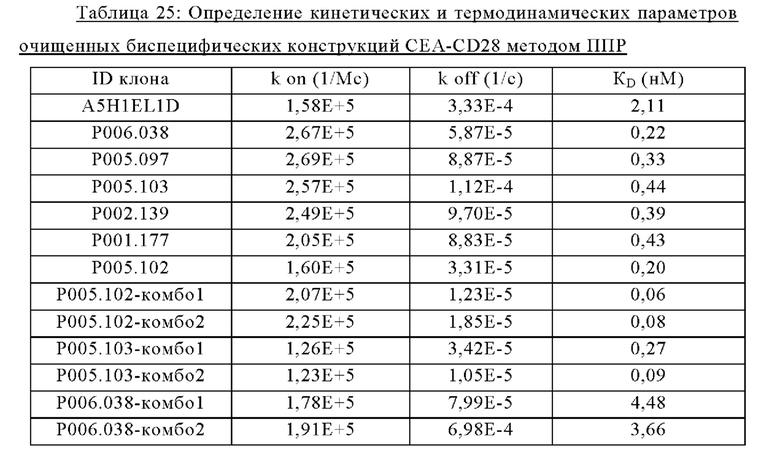
Пример 11
Создание и выработка дополнительных биспецифических антигенсвязывающих молекул, нацеленных на CD28 и карциноэмбриональный антиген (СЕА)
11.1 Клонирование биспецифических антигенсвязывающих молекул
Для создания экспрессионных плазмид использовали последовательности соответствующих вариабельных доменов, которые субклонировали в рамке с соответствующими константными областями, которые были предварительно вставлены в соответствующий реципиентный экспрессионный вектор млекопитающих. Схематическое описание полученных в результате молекул приведено на Фиг. 23. В Fc-домене мутации Pro329Gly, Leu234Ala и Leu235Ala (PG-LALA) были внесены в константную область тяжелых цепей IgG1 человека для устранения связывания с Fc-гамма-рецепторами в соответствии с методом, описанным в публикации заявки на Международный патент № WO 2012/130831. Для создания биспецифических антител Fc-фрагменты содержали мутации типа «выступ» (мутации 8354С/T366W, нумерация в соответствии с индексом EU по Kabat) или типа «впадина» (мутации Y349C/T366S/L368A/Y407V, нумерация в соответствии с индексом EU по Kabat) для предотвращения неправильного спаривания тяжелых цепей. Чтобы предотвратить неправильное спаривание легких цепей в биспецифических антигенсвязывающих молекулах, в один связывающий фрагмент вносили обмен доменов VH/VL или СН1/С-каппа (технология CrossFab). В другом связывающем фрагменте в домены СН1 и С-каппа вносили заряды, как описано в публикации заявки на Международный патент № WO 2015/150447. Создание и подготовка анти-СЕА клона Т84.66 описаны в WO 2016/075278 А2.
Клонировали следующие молекулы, схематические иллюстрации которых показаны на Фиг. 23А-23Г:
Молекула 11А: CEA (A5H1EL1D)-CD28 (SA) формата 1+1, биспецифическая молекула huIgG1 PG-LALA CrossFab с заряженной модификацией в Fab-фрагменте (выступ) CD28(SA) и обменом VH/VL в Fab-фрагменте (впадина) CEA(A5F£1EL1D) (Фиг. 23А), содержащая аминокислотные последовательности SEQ ID NO: 351, 352, 353 и 354 (Р1АЕ4773).
Молекула 11В: CEA (A5H1EL1D)-CD28 (SA_вариант 8) формата 1+1, биспецифическая молекула huIgG1 PG-LALA CrossFab с заряженной модификацией в Fab-фрагменте (выступ) CD28(SA_вариант 8) и обменом VH/VL в Fab-фрагменте (впадина) CEA(A5H1EL1D) (Фиг. 23А), содержащая аминокислотные последовательности SEQ ID NO: 351, 352, 355 и 356 (Р1АЕ4774).
Молекула 11С: CEA (A5H1EL1D)-CD28 (SA_вариант 15) формата 1+1, биспецифическая молекула huIgG1 PG-LALA CrossFab с заряженной модификацией в Fab-фрагменте (выступ) CD28(SA вариант 15) и обменом VH/VL в Fab-фрагменте (впадина) CEA(A5H1EL1D) (Фиг. 23А), содержащая аминокислотные последовательности SEQ ID NO: 351, 352, 357 и 358 (Р1АЕ4775).
Молекула 11D: СЕА (A5H1EL1D)-CD28 (SA_вариант 29) формата 1+1, биспецифическая молекула huIgG1 PG-LALA CrossFab с заряженной модификацией в Fab-фрагменте (выступ) CD28(SA_вариант 29) и обменом VH/VL в Fab-фрагменте (впадина) CEA(A5H1EL1D) (Фиг. 23А), содержащая аминокислотные последовательности SEQ ID NO: 351, 352, 359 и 354 (Р1АЕ4776).
Молекула 11Е: СЕА (A5H1EL1D)-CD28 (SA) формата 1+1, биспецифическая молекула huIgG1 PG-LALA CrossFab с обменом VH/VL в Fab-фрагменте (выступ) CD28(SA) и заряженными модификациями в Fab-фрагменте (впадина) CEA(A5H1EL1D) (Фиг. 23Б), содержащая аминокислотные последовательности SEQ ID NO: 360, 361, 362 и 363 (Р1АЕ4777).
Молекула 11F: СЕА (A5H1EL1D)-CD28 (SA_вариант 8) формата 1+1, биспецифическая молекула huIgG1 PG-LALA CrossFab с обменом VH/VL в Fab-фрагменте (выступ) CD28 (SA_вариант 8) и заряженными модификациями в Fab-фрагменте (впадина) CEA(A5H1EL1D) (Фиг. 23Б), содержащая аминокислотные последовательности SEQ ID NO: 360, 361, 364 и 365 (Р1АЕ4780).
Молекула 11G: СЕА (A5H1EL1D)-CD28 (SA_вариант 15) формата 1+1, биспецифическая молекула huIgG1 PG-LALA CrossFab с обменом VH/VL в Fab-фрагменте (выступ) CD28 (SA вариант 15) и заряженными модификациями в Fab-фрагменте (впадина) CEA(A5H1EL1D) (Фиг. 23Б), содержащая аминокислотные последовательности SEQ ID NO: 360, 361, 366 и 367 (Р1АЕ4791).
Молекула 11Н: СЕА (A5H1EL1D)-CD28 (SA_вариант 29) формата 1+1, биспецифическая молекула huIgG1 PG-LALA CrossFab с обменом VH/VL в Fab-фрагменте (выступ) CD28 (SA_вариант 29) и заряженными модификациями в Fab-фрагменте (впадина) CEA(A5H1EL1D) (Фиг. 23Б), содержащая аминокислотные последовательности SEQ ID NO: 360, 361, 368 и 363 (Р1АЕ4793).
Молекула 11I: СЕА (T84.66)-CD28 (SA) формата 1+1, биспецифическая молекула huIgG1 PG-LALA CrossFab с заряженной модификацией в Fab-фрагменте (выступ) CD28(SA) и обменом VH/VL в Fab-фрагменте (впадина) СЕА(Т84.66) (Фиг. 23А), содержащая аминокислотные последовательности SEQ ID NO: 369, 370, 353 и 354 (Р1АЕ6488).
Молекула 11J: СЕА (T84.66)-CD28 (SA_вариант 29) формата 1+1, биспецифическая молекула huIgGl PG-LALA CrossFab с заряженной модификацией в Fab-фрагменте (выступ) CD28(SA_вариант 29) и обменом VH/VL в Fab-фрагменте (впадина) СЕА(Т84.66) (Фиг. 23А), содержащая аминокислотные последовательности SEQ ID NO: 369, 370, 359 и 354 (Р1АЕ6495).
Молекула 11K: СЕА (T84.66)-CD28 (SA_вариант 15) формата 1+1, биспецифическая молекула huIgG1 PG-LALA CrossFab с заряженной модификацией в Fab-фрагменте (выступ) CD28(SA вариант 15) и обменом VH/VL в Fab-фрагменте (впадина) СЕА(Т84.66) (Фиг. 23А), содержащая аминокислотные последовательности SEQ ID NO: 369, 370, 357 и 358 (Р1АЕ6557).
Молекула 11L: СЕА (T84.66)-CD28 (SA_вариант 8) формата 1+1, биспецифическая молекула huIgG1 PG-LALA CrossFab с заряженной модификацией в Fab-фрагменте (выступ) CD28(SA_вариант 8) и обменом VH/VL в Fab-фрагменте (впадина) СЕА(Т84.66) (Фиг. 23А), содержащая аминокислотные последовательности SEQ ID NO: 369, 370, 355 и 356 (Р1АЕ6556).
Молекула ИМ: СЕА (T84.66)-CD28 (SA_вариант 29) формата 1+1, биспецифическая молекула huIgG1 PG-LALA CrossFab с обменом VH/VL в Fab-фрагменте (выступ) CD28(SA_вариант 29) и заряженными модификациями в Fab-фрагменте (впадина) СЕА(Т84.66) (Фиг. 23Б), содержащая аминокислотные последовательности SEQ ID NO: 371, 372, 368 и 363 (Р1АЕ9605).
Молекула 11N: СЕА (T84.66)-CD28 (SA_вариант 8) формата 1+1, биспецифическая молекула huIgG1 PG-LALA CrossFab с обменом VH/VL в Fab-фрагменте (выступ) CD28 (SA_вариант 8) и заряженными модификациями в Fab-фрагменте (впадина) СЕА(Т84.66) (Фиг. 23Б), содержащая аминокислотные последовательности SEQ ID NO: 371, 372, 366 и 367 (Р1АЕ9606).
Молекула 11О: СЕА (T84.66)-CD28 (SA_вариант 15) формата 1+1, биспецифическая молекула huIgG1 PG-LALA CrossFab с обменом VH/VL в Fab-фрагменте (выступ) CD28 (вАвариант 15) и заряженными модификациями в Fab-фрагменте (впадина) СЕА(Т84.66) (Фиг. 23Б), содержащая аминокислотные последовательности SEQ ID NO: 371, 372, 364 и 365 (Р1АЕ9607).
Молекула 11P: СЕА (A5H1EL1D)-CD28 (SA-вариант 29) 2+1, биспецифическая одновалентная анти-CD28 (SA_вариант 29) и двухвалентная анти-СЕА huIgG1 PG-LALA кросс-Fab конструкция, заряженные модификации в обоих-СЕА Fab-фрагментах, слитых «голова к хвосту» друг с другом (впадина), обмен VH/VL в анти-CD28 Kpocc-Fab-фрагменте (выступ) (Фиг. 23В). Молекула содержит аминокислотные последовательности из SEQ ID NO: 368, 362, 361 и 373 (Р1АЕ6924).
Молекула 11Q: СЕА (A5H1EL1D)-CD28 (SA_вариант 29) 2+1, биспецифическая одновалентная анти-CD28 (SA) и двухвалентная анти-СЕА huIgG1 PG-LALA конструкция кросс-Fab, «классическая ориентация», обмен VH/VL в анти-CD28 Kpocc-Fab-фрагменте, заряженная модификация в обоих анти-СЕА Fab-фрагментах (Фиг. 23Г). Молекула содержит аминокислотные последовательности из SEQ ID NO: 368, 374, 361 и 360 (Р1АЕ6925).
Молекула 11R: СЕА (Р002.139)-CD28 (8А_вариант 15) формата 1+1, биспецифическая молекула huIgG1 PG-LALA CrossFab с заряженной модификацией в Fab-фрагменте (выступ) CD28(SA_вариант 15) и обменом VH/VL в Fab-фрагменте (впадина) СЕА(Р002.139) (Фиг. 23А), содержащая аминокислотные последовательности SEQ ID NO: 375, 376, 357 и 358 (Р1АЕ8371).
Молекула 11S: СЕА (Р002.139)-CD28 (SA_вариант 8) формата 1+1, биспецифическая молекула huIgG1 PG-LALA CrossFab с заряженной модификацией в Fab-фрагменте (выступ) CD28(SA_вариант 8) и обменом VH/VL в Fab-фрагменте (впадина) СЕА(Р002.139) (Фиг. 23А), содержащая аминокислотные последовательности SEQ ID NO: 375, 376, 355 и 356 (P1AF1115).
Молекула 11Т: СЕА (Р002.139)-CD28 (SA_вариант 11) формата 1+1, биспецифическая молекула huIgG1 PG-LALA CrossFab с заряженной модификацией в Fab-фрагменте (выступ) CD28(SA_вариант 11) и обменом VH/VL в Fab-фрагменте (впадина) СЕА(Р002.139) (Фиг. 23А), содержащая аминокислотные последовательности SEQ ID NO: 375, 376, 355 и 354 (P1AF1116).
Для сравнения были созданы следующие варианты анти-CD28 антитела:
Молекула 11U: CD28(SA_вариант 8) (PG-LALA), CD28 (SA_вариант 8) антитело в изотипе huIgG1 PG-LALA (Фиг. 1Б) содержит аминокислотные последовательности SEQ ID NO: 377 и SEQ ID NO: 378 (P1AE7035).
Молекула 11V: CD28(SA_вариант 11) (PG-LALA), CD28 (SA_вариант 11) антитело в изотипе huIgG1 PG-LALA (Фиг. 1Б) содержит аминокислотные последовательности SEQ ID NO: 379 и SEQ ID NO: 380 (Р1АЕ7036).
Молекула 11W: CD28(SA_вариант 15) (PG-LALA), CD28 (SA_вариант 15) антитело в изотипе huIgG1 PG-LALA (Фиг. 1Б) содержит аминокислотные последовательности SEQ ID NO: 381 и SEQ ID NO: 382 (P1AE7037).
Молекула 11Х: CD28(SA_вариант 27) (PG-LALA), CD28 (SA_вариант 27) антитело в изотипе huIgG1 PG-LALA (Фиг. 1Б) содержит аминокислотные последовательности SEQ ID NO: 383 и SEQ ID NO: 384 (P1AE7038).
Молекула 11Y: С D2 8 (SA_вариант 29) (PG-LALA), CD28 (SA_вариант 29) антитело в изотипе huIgG1 PG-LALA (Фиг. 1Б) содержит аминокислотные последовательности SEQ ID NO: 385 и SEQ ID NO: 386 (P1AE7039).
11.2 Выработка молекул
Экспрессией вышеуказанных молекул управляет химерный промотор MPSV или промотор CMV. Полиаденилирование обусловлено синтетической последовательностью сигнала поли-А, расположенной в 3' конце КП. Кроме того, каждый вектор содержит последовательность EBV OriP для аутосомальной репликации.
Для выработки молекул 11A-11D и 11I-11T, клетки HEK293-EBNA, которые росли в суспензии, котрансфицировали соответствующими экспрессионными векторами, используя в качестве трансфекционного реагента полиэтиленимин. Антитела и биспецифические антитела создавали с помощью временной трансфекции клеток НЕК293 EBNA. Клетки центрифугировали и заменяли среду предварительно нагретой средой CD СНО. Экспрессионные векторы смешивали в среде CD СНО, добавляли ПЭИ, перемешивали раствор на вортексе и инкубировали 10 минут при комнатной температуре. После этого клетки смешивали с раствором ДНК/ПЭИ, переносили во встряхиваемую колбу и инкубировали 3 часа при 37°С в инкубаторе с атмосферой 5% СО2. После инкубации добавляли среду Excell с добавками (культуры клеток млекопитающих для производства биопрепаратов, редакторы: Weichang Zhou, Anne Kantardjieff). Через одни сутки после трансфекции добавляли добавки (Feed) (культуры клеток млекопитающих для производства биопрепаратов, редакторы: Weichang Zhou, Anne Kantardjieff). Клеточные супернатанты собирали через 7 суток путем центрифугирования и последующей фильтрации (0,2 мкм фильтр) и очищали стандартными методами.
Молекулы 11U, 11V, 11W, 11Х и 11Y были получены и очищены Evitria с использованием их собственной векторной системы с помощью стандартных (не на основе ГЩР) методик клонирования и использованием адаптированных к суспензии клеток СНО K1 (изначально полученных от АТСС и адаптированных для бессывороточного роста в суспензионной культуре в Evitria). Для выработки Evitria использовали их собственные не содержащую животных компонентов и не содержащую сыворотку среды (eviGrow и eviMake2) и их собственный трансфекционный реагент (eviFect). Супернатант собирали путем центрифугирования и последующей фильтрации (0,2 мкм фильтр) и очищали стандартными методами. Молекулы 11Е, 11F, 11G и 11Н были получены и очищены Proteros в соответствии с их стандартными методами и протоколами.
11.3 Очистка молекул
Белки очищали из профильтрованных клеточных культуральных супернатантов согласно стандартным протоколам. Вкратце, Fc-содержащие белки очищали из клеточных культуральных супернатантов с помощью аффинной хроматографии с протеином А (уравновешивающий буфер: 20 мМ цитрат натрия, 20 мМ фосфат натрия, рН 7,5; элюирующий буфер: 20 мМ цитрат натрия, рН 3,0). Элюирование проводили при рН 3,0 с последующей незамедлительной нейтрализацией рН образца. Белок концентрировали путем центрифугирования (Millipore Amicon® ULTRA-15 (Изд. №: UFC 903096) и отделяли агрегированный белок от мономерного белка с помощью эксклюзионной хроматографии в 20 мМ гистидине, 140 мМ хлориде натрия, рН 6,0.
11.4 Аналитические данные по биспецифическим антигенсвязывающим молекулам, нацеленным на CD28 и карциноэмбриональный антиген (СЕА)
Концентрацию очищенных белков определяли путем измерения поглощения на 280 нм, используя массовый коэффициент экстинкции, рассчитанный на основании аминокислотной последовательности, в соответствии с Расе, et al., Protein Science, 1995, 4, 2411-1423. Чистоту и молекулярную массу белков анализировали с помощью КЭ-ДСН в присутствии или отсутствие восстанавливающего агента, используя LabChipGXII (Perkin Elmer). Определение содержания агрегатов проводили с помощью ВЭЖХ-хроматографии при 25°С, используя аналитическую эксклюзионную колонку (TSKgel G3000 SW XL или UP-SW3000), уравновешенную в подвижном буфере (25 мМ K2HPO4, 125 мМ NaCl, 200 мМ L-аргинина моногидрохлорида, рН 6,7, или 200 мМ KH2PO4, 250 мМ KCl, рН 6,2, соответственно). Обобщенные данные по параметрам очистки всех молекул приведены в таблице 26.
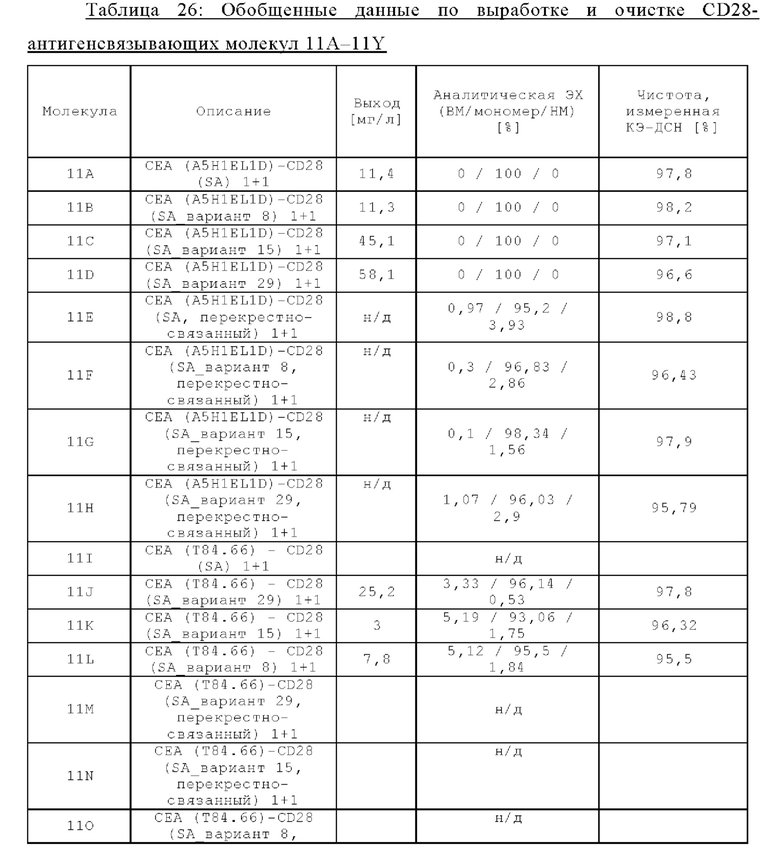
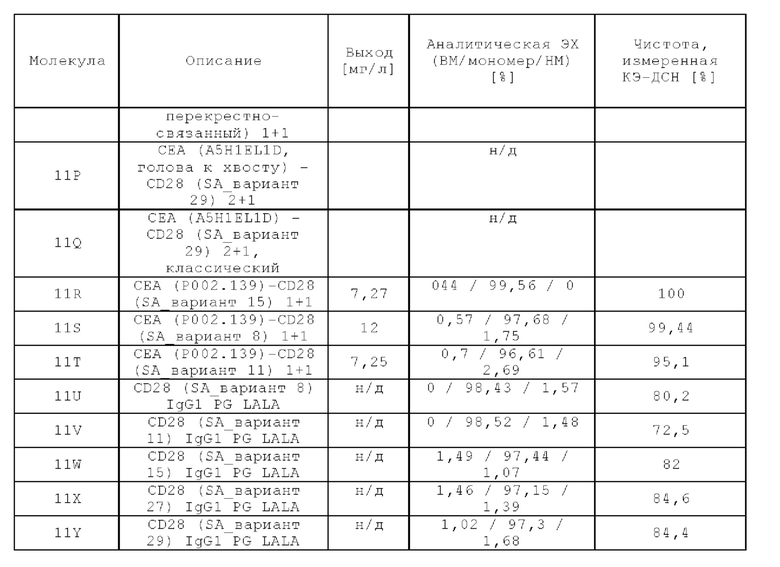
11.5 Анализ связывания биспецифических антигенсвязывающих молекул, нацеленных на CD28 и карциноэмбриональный антиген (СЕА) методом ППР
Аффинность (KD) к CD28 молекул 10A-10D, которые несут анти-СЕА антитело A5H1EL1D и анти-CD28 связывающие варианты 8, 11 и 29, а также оригинальную связывающую молекулу CD28(SA), измеряли методом поверхностного плазмонного резонанса или ППР, используя прибор Proteon XPR36. Для иммобилизации рекомбинантного антигена (лиганда) huCD28-Fc разводили ФСБТ (10 мМ фосфата, 150 мМ хлорида натрия, рН 7,4, 0,005% Твин 20), до концентрации в диапазоне от 100 до 500 нМ, потом вводили при 25 мкл/минута с переменным временем контакта. Это приводило к уровням иммобилизации от 500 до 3000 единиц ответа (ЕО) в вертикальной ориентации.
Для определения аффинности (KD) очищенных молекул направление введения меняли на горизонтальную ориентацию. Двукратные серийные разведения очищенных конструкций (диапазон переменных концентраций от 100 до 6,25 нМ) вводили одновременно при 100 мкл/мин вдоль отдельных каналов 1-5 со временем ассоциации 150 с и временем диссоциации 400 с. Буфер (ФСБТ) вводили вдоль шестого канала для обеспечения «параллельной» холостой пробы для сравнения. Восстановление проводили 10 мМ глицина, рН 1,5, в течение 35 с при 50 μл/мин (вертикальная ориентация). Константы скорости ассоциации (kon) и константы скорости диссоциации (koff) рассчитывали, используя простую один-к-одному модель связывания Лэнгмюра в программном обеспечении ProteOn Manager v3.1, путем одновременной аппроксимации сенсограмм ассоциации и диссоциации. Равновесную константу диссоциации (KD) рассчитывали как соотношение koff/kon. Кинетические и термодинамические данные приведены в таблице 27.
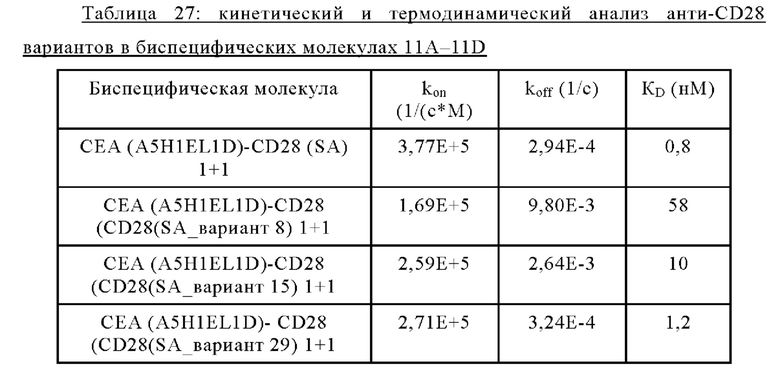
11.6 Биохимические характеристики анти-CD28 вариантов IgG и отобранных 1+1 биспецифических нацеленных на СЕА анти-CD28 антигенсвязывающих молекул
Чтобы получить характеристики и сравнить их биохимические и биофизические свойства, подробно анализировали следующие молекулы: Нацеленные на СЕА анти-CD28 биспецифические варианты, молекулы 10A-10D, м анти-CD28 IgG варианты, молекулы 10U-10Y. Результаты обобщены в Таблицах 27 и 28.
Хроматография с гидрофобным взаимодействием (HIC)
Кажущуюся гидрофобность определяли путем введения 20 мкг образца в колонку HIC-Ether-5PW (Tosoh), уравновешенную 25 мМ Na-фосфата, 1,5 М сульфата аммония, рН 7,0. Элюирование проводили линейным градиентом от 0 до 100% буфера В (25 мМ Na-фосфат, рН 7,0) за 60 минут. Время удержания сравнивали с белковыми стандартами с известной гидрофобностью. Большинство антител демонстрировали относительное время удержания от 0 до 0,35.
Термостабильность
Образцы готовили в концентрации 1 мг/мл в 20 мМ смеси гистидина/хлорида гистидина, 140 мМ NaCl, рН 6,0, переносили в оптический 384-луночный планшет путем центрифугирования через 0,4 мкм фильтровальную пластину и покрывали парафинным маслом. Гидродинамический радиус измеряли повторно методом динамического рассеяния света на планшет-ридере DynaPro (Wyatt), в то время как образцы нагревали при скорости 0,05°С/мин от 25°С до 80°С.
Аффинная хроматография FcRn
FcRn экспрессировали, очищали и биотинилировали, как описано в Cymer, Schlothauer et al., Bioanalysis 2017, 9(17), doi.org/10.4155/bio-2017-0109. Для сопряжения полученный рецептор добавляли к стрептавидин-сефарозе (GE Healthcare). Полученную в результате матрицу FcRn-сефароза упаковывали в кожух колонки. Колонку уравновешивали 20 мМ 2-(N-морфолин)-этансульфоновой кислотой (МЭС), 140 мМ NaCl, рН 5,5 (элюент А) при скорости потока 0,5 мл/мин. 30 мкг образцов антитела разводили в объемном соотношении 1:1 элюентом А и наносили на колонку с FcRn. Колонку промывали 5 объемами колонки элюента А с последующим элюированием линейным градиентом от 20 до 100% 20 мМ Трис/HCl, 140 мМ NaCl, рН 8,8 (элюент В) в 35 объемах колонки. Анализ проводили с колоночным термостатом при 25°С.Профиль элюирования отслеживали путем непрерывного измерения поглощения на 280 нм. Время удержания сравнивали с белковыми стандартами с известной аффинностью. Большинство антител демонстрировали относительное время удержания от 0 до 1.
Гепариновая аффинная хроматография
Аффинность гепарина определяли путем введения 30 50 мкг образца в колонку TSKgel Heparin-5PW (Tosoh), уравновешенную 50 мМ Трис, рН 7,4. Элюирование проводили линейным градиентом от 0 до 100% буфера В (50 мМ Трис, 1 М NaCl, рН 7,4) за 37 минут. Время удержания сравнивали с белковыми стандартами с известной аффинностью.
Все исследованные варианты по последовательности удовлетворяли всем критериям и существенно не отличались друг от друга в отношении всех исследуемых биофизических и биохимических свойств (таблицы 28 и 29).
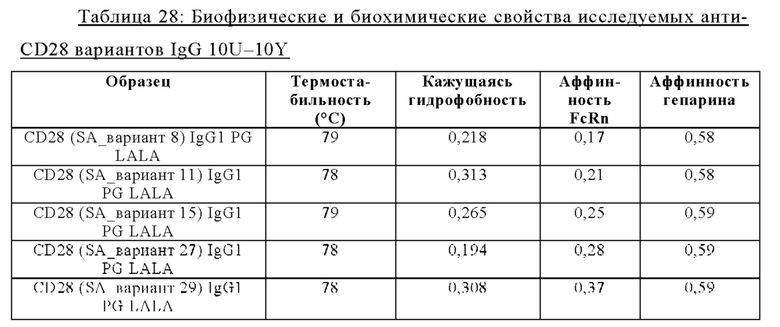

Пример 12
In vitro функциональные характеристики биспецифических антигенсвязывающих молекул, нацеленных на CD28 и карциноэмбриональный антиген (СЕА)
12.1 Связывание с СЕАСАМ5 на экспрессирующих СЕА клетках MV3
Чтобы оценить, привело ли созревание аффинности A5H1EL1D к улучшению связывания с СЕАСАМ5, проводили анализ связывания FACS на клетках MV3, генетически модифицированных, чтоб экспрессировать СЕА. Как показано на Фиг. 24, биспецифическое антитело CEA-CD28 (молекула 11R, Р1АЕ8371), несущее анти-СЕА клон Р002.139 с созревшей аффинностью, демонстрировало превосходящее связывание (ЕС50 = 4,1 нМ) с СЕАСАМ5 по сравнению с клоном A5H1EL1D (молекула ПС, Р1АЕ4775) (ЕС50 = 20,4 нМ).
12.2 Репортерный анализ с IL-2 для анализа in vitro функциональности биспецифических антител к CEA-CD28 в комбинации с нацеленными на СЕА ТСВ
Репортерные клетки IL-2 (Л 631, Promega) представляют собой генетически сконструированные Т-клетки Jurkat, которые экспрессируют репортер люциферазы, управляемый промотором IL-2. Чтобы оценить способность нацеленных на СЕА агонистов CD28 усиливать ТСВ-опосредованную эффекторную функцию Т-клеток, 10000 клеток MKN45/лунка инкубировали с 105 репортерных клеток IL-2 (соотношение Э:М 10:1) с фиксированной концентрацией СЕА-ТСВ (5 нМ) и диапазоном концентраций биспецифических антител к CEA-CD28 (14 пМ - 10 нМ). Через 6 ч инкубации при 37°С, 5% CO2, оценивали люминесценцию, используя OneGlo (Е6120, Promega) в соответствии с инструкциями производителя. Планшеты считывали на планшет-ридере Tecan Spark 10 М.
Функциональность биспецифического антитела к CEA-CD28, несущего анти-СЕА клон Р002.139 с созревшей аффинностью (молекула 11R, Р1АЕ8371) или клон A5H1EL1D (молекула 11С, Р1АЕ4775), оценивали, используя анализ с репортерными клетками IL-2 в комбинации с СЕА-ТСВ (5 нМ) и в присутствии экспрессирующих СЕА клеток MKN45. Как показано на Фиг. 25А и 25Б, улучшенная аффинность клона Р002.139 с созревшей аффинностью транслировалась в более высокую активность в отношении костимулирующей способности по сравнению с клоном A5H1EL1D.
Пример 13
In vivo функциональные характеристики биспецифических антигенсвязывающих молекул, нацеленных на CD28 и карциноэмбриональный антиген (СЕА), в комбинации с СЕА ТСВ
13.1 Исследование эффективности биспецифических антигенсвязывающих молекул к CEA-CD28 с разными СЕА-антигенсвязывающими доменами в комбинации с СЕА-ТСВ в ксенотрансплантате MKN45 у гуманизированных мышей Целью описанного в данном документе исследования эффективности было понять зависимую от СЕА-клона активность биспецифической антигенсвязывающей молекулы к CEA-CD28 в комбинации с СЕА-ТСВ в терминах регрессии опухоли в полностью гуманизированных мышей NSG.
Человеческие клетки MKN45 (карцинома желудка человека) были изначально получены от АТСС и после размножения помещены во внутренний банк клеток Glycart. Клетки культивировали в DMEM, содержащей 10% ФТС, при 37°С в насыщенной влагой атмосфере при 5% СО2. Для подкожной инъекции использовали in vitro пассаж 12 при жизнеспособности 97%. 50 микролитров клеточной суспензии (1×106 клеток MKN45), смешанной с 50 микролитрами матригеля, подкожно инъецировали в бок анестезированных мышей с помощью 22G-30G иглы.
Самок мышей NSG возрастом 4 5 недель на момент начала эксперимента (Jackson Laboratory) содержали в специальных беспатогенных условиях с суточными циклами 12 ч день/12 ч ночь в соответствии с принятыми руководствами (GV-Solas; Felasa; TierschG). Протокол экспериментального исследования был рассмотрен и одобрен местными органами управления (ZH225-17). После поступления животных держали в течение одной недели для привыкания к новой среде и для наблюдения. Постоянное наблюдение за состоянием здоровья осуществляли на регулярной основе.
Самкам мышей NSG вводили в/б 15 мг/кг бусульфана, за чем через день следовала в/в инъекция 1×105 человеческих гемопоэтических клеток, выделенных из пуповинной крови. На 14-16 неделе после инъекции стволовых клеток у мышей сублингвально брали кровь и анализировали ее методом проточной цитометрии в отношении успешной гуманизации. Мышей с эффективным прививанием рандомизировали в соответствии с частотой человеческих Т-клеток в разные группы обработки. В это время мышам вводили п/к опухолевые клетки, как описано (Фиг. 26, с0) и обрабатывали соединениями или гистидиновым буфером (носитель), когда размер опухолей достигал прибл. 150 мм3 (сутки 13). Всем мышам вводили в/в 200 мкл соответствующего раствора. Для получения надлежащего количества соединений на 200 мкл исходные растворы (таблица 30) разводили при необходимости гистидиновым буфером.
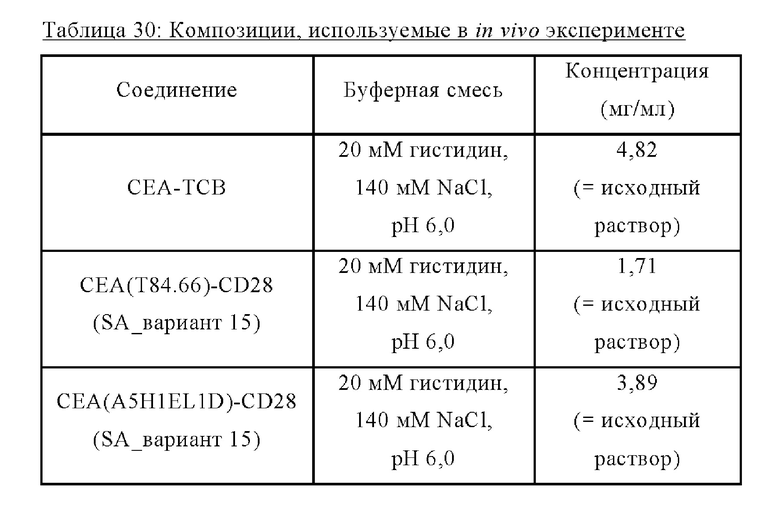
В случае комбинированной терапии (группа В и Г, Фиг. 26) CEA-CD28 и биспецифические молекулы СЕА-ТСВ вводили одновременно. Рост опухоли измеряли дважды в неделю, используя штангенциркуль, а объем опухолей рассчитывали следующим образом:

Ингибирование роста опухоли, как показатель активности in vivo, рассчитывали с помощью внутренне статистической программы Roche следующим образом:
Исследование заканчивали на 39 сутки. На Фиг. 27А показана кинетика роста опухоли (среднее + СПС), а также индивидуальная кинетика роста опухоли на группу и на мышь (Фиг. 27Б-27Д). Как описано в данном документе, СЕА-ТСВ как отдельный агент индуцировал слабое ингибирование роста опухолей. Однако комбинации с обеими молекулами CEA-CD28 демонстрировали улучшение ингибирования роста опухолей. В особенности комбинация СЕА-ТСВ с CEA-CD28, которая содержит связывающую СЕА молекулу с меньшей аффинностью (A5H1EL1D), приводила к превосходящей регрессии роста опухолей. Как показано в таблице 31, наибольшее значение ИРО (ИРО: 116%) было рассчитано для комбинированной группы СЕА-ТСВ с СЕА-CD28 (huA5B7), свидетельствуя о самых сильных противоопухолевых эффектах. ИРО означает ингибирование роста опухоли, ИРО > 100 означает регрессию опухоли, а ИРО = 100 определяется как стазис опухоли.
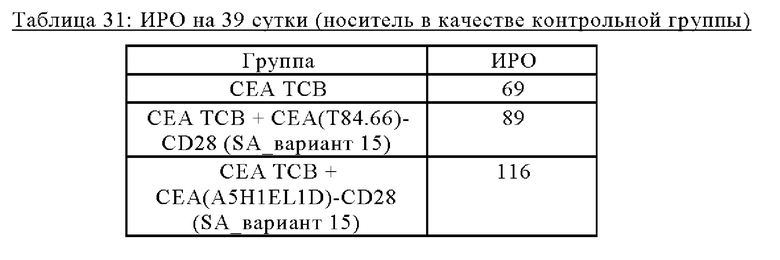
13.2 Исследование эффективности биспецифических антигенсвязывающих молекул к CEA-CD28 с тремя разными CD28-антигенсвязывающими доменами в комбинации с СЕАСАМ5-ТСВ и анти-PDL1 антителом в ксенотрансплантате ВХРС3 у гуманизированных мышей
Целью этого исследования эффективности было понимание влияния аффинности СВ28-антигенсвязывающего домена молекул CEA-CD28 в комбинации с СЕАСАМ5-ТСВ и анти-PD-L1 в терминах регрессии опухоли и иммуно-ФД профилей у полностью гуманизированных мышей NSG. В данном исследовании тестировали три варианта с разной аффинностью (вариант 8 < вариант 15 < вариант 29).
Человеческие клетки ВХРС3 (линия клеток рака поджелудочной железы человека) были изначально получены от ЕСАСС (Европейская коллекция клеточных культур) и после размножения помещены во внутренний банк клеток Roche Glycart. Клетки ВХРС3 культивировали в RPMI, содержащей 10% ФТС (РАА Laboratories, Austria) с 1% Glutamax. Клетки культивировали при 37°С в насыщенной влагой атмосфере при 5% СО2. Для и/к инъекции использовали in vitro пассаж 20 при жизнеспособности > 95%. 50 микролитров клеточной суспензии (1×106 клеток ВХРС3), смешанной с 50 микролитрами матригеля, подкожно инъецировали в бок анестезированных мышей с помощью 22G-30G иглы.
Самок мышей NSG возрастом 4-5 недель на момент начала эксперимента (Jackson Laboratory) содержали в специальных беспатогенных условиях с суточными циклами 12 ч день/12 ч ночь в соответствии с принятыми руководствами (GV-Solas; Felasa; TierschG). Протокол экспериментального исследования был рассмотрен и одобрен местными органами управления (ZH225-17). После поступления животных держали в течение одной недели для привыкания к новой среде и для наблюдения. Постоянное наблюдение за состоянием здоровья осуществляли на регулярной основе.
Самкам мышей NSG вводили в/б 15 мг/кг бусульфана, за чем через день следовала в/в инъекция 1×105 человеческих гемопоэтических клеток, выделенных из пуповинной крови. На 14-16 неделе после инъекции стволовых клеток у мышей сублингвально брали кровь и анализировали ее методом проточной цитометрии в отношении успешной гуманизации. Мышей с эффективным прививанием рандомизировали в соответствии с частотой человеческих Т-клеток в разные группы обработки от А до Е. В это время мышам воодили п/к опухолевые клетки, как описано на Фиг. 28, и обрабатывали соединениями или гистидиновым буфером (носитель), когда размер опухолей достигал прибл. 150 мм3 (сутки 20). Всем мышам вводили в/в 200 мкл соответствующего раствора. Для получения надлежащего количества соединений на 200 мкл исходные растворы (таблица 32) разводили при необходимости гистидиновым буфером.
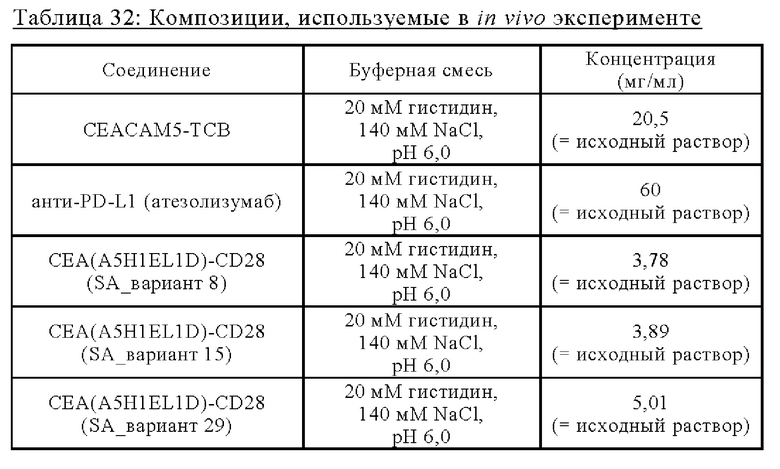
В конце исследования (сутки 52) мышей умерщвляли, удаляли опухоли, взвешивали и готовили суспензии из одиночных клеток путем ферментативного расщепления коллагеназой V и ДНКазой для последующего анализа FACS.
Одиночные клетки окрашивали в отношении CD45, CD3, CD8 и CD4 человека и анализировали на FACS BDFortessa.
На Фиг. 29 показана кинетика роста опухолей (среднее, + СПС) для всех групп обработки, соответствующие значения ИРО для каждой группы обработки приведены в таблице 33 ниже. Как описано в данном документе, монотерапия СЕАСАМ5 ТСВ, а также комбинация с a-PD-L1 индуцировали слабое ингибирование роста опухолей. Только добавление CEA-CD28 (SA_вариант 8) к комбинации СЕАСАМ5-ТСВ и a-PD-L1 привело к повышению ингибирования роста опухолей (ИРО: 75%). Ни вариант 15, ни вариант 29 не повышали противоопухолевые эффекты. Что интересно, данные иммуно-ФД (Фиг. 30 А-Г) по опухолям от животных, умерщвленных на момент завершения исследования, показали, что дополнительное ингибирование роста опухолей, индуцированное вариантом 8 CEA-CD28, также отображается в повышении частоты Т-клеток внутри опухоли. На Фиг. 30 А приведены репрезентативные точечные графики окрашенных суспензий одиночных опухолевых клеток для каждой группы обработки. Обобщенные данные по инфильтрации CD3, CD8 и CD4 Т-клеток приведены на Фиг. 30Б, 30В и 30Г, соответственно. В группах, обработанных CEA-CD28 (SA_вариант 15) или CEA-CD28 (SA_вариант 29) по сравнению с одним СЕАСАМ5-ТСВ или комбинацией с a-PD-L1 не наблюдали статистической разницы в терминах инфильтрации Т-клеток в опухоль. Наиболее сильные иммуно-ФД эффекты были выявлены с CEA-CD28 (SA_вариант 8).
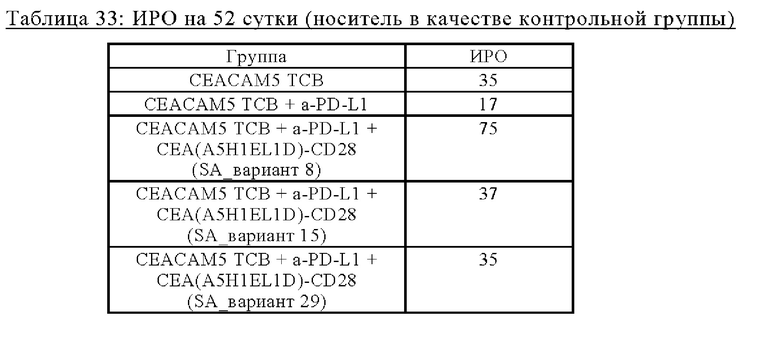
13.3 Исследование эффективности биспецифической антигенсвязывающей молекулы к CEA-CD28 в комбинации с СЕА-ТСВ в ксенотрансплантате MKN45 у гуманизированных мышей
Целью описанного в данном документе исследования эффективности было исследование комбинации СЕА-ТСВ и CEA-CD28 (SA_вариант 8) во второй модели с человеческим ксенотрансплантатом в терминах регрессии опухоли и профилей иммуно-ФД у полностью гуманизированных мышей NSG.
Человеческие клетки MKN45 (карцинома желудка человека) были изначально получены от АТСС и после размножения помещены во внутренний банк клеток Glycart. Клетки культивировали в DMEM, содержащей 10% ФТС, при 37°С в насыщенной влагой атмосфере при 5% CO2. Для подкожной инъекции использовали in vitro пассаж 12 при жизнеспособности > 95%. 50 микролитров клеточной суспензии (1×106 клеток MKN45), смешанной с 50 микролитрами матригеля, подкожно инъецировали в бок анестезированных мышей с помощью 22G-30G иглы. Самок мышей NSG возрастом 4 5 недель на момент начала эксперимента (Jackson Laboratory) содержали в специальных беспатогенных условиях с суточными циклами 12 ч день/12 ч ночь в соответствии с принятыми руководствами (GV-Solas; Felasa; TierschG). Протокол экспериментального исследования был рассмотрен и одобрен местными органами управления (ZH225-17). После поступления животных держали в течение одной недели для привыкания к новой среде и для наблюдения. Постоянное наблюдение за состоянием здоровья осуществляли на регулярной основе.
Самкам мышей NSG вводили в/б 15 мг/кг бусульфана, за чем через день следовала в/в инъекция 1x105 человеческих гемопоэтических клеток, выделенных из пуповинной крови. На 14 16 неделе после инъекции стволовых клеток у мышей сублингвально брали кровь и анализировали ее методом проточной цитометрии в отношении успешной гуманизации. Мышей с эффективным прививанием рандомизировали в соответствии с частотой человеческих Т-клеток в разные группы обработки. В это время мышам вводили п/к опухолевые клетки, как описано на Фиг. 31, и обрабатывали молекулами или гистидиновым буфером (носитель), когда размер опухолей достигал прибл. 150 мм3 (сутки 13). Всем мышам вводили в/в 200 мкл соответствующего раствора.
Для получения надлежащего количества соединений на 200 мкл исходные растворы (таблица 34) разводили при необходимости гистидиновым буфером.
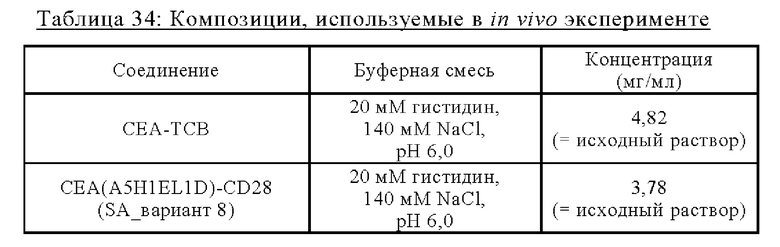
В случае комбинированной терапии (группа В, Фиг. 31) конструкции вводили одновременно. Рост опухоли измеряли дважды в неделю, используя штангенциркуль, а объем опухолей рассчитывали следующим образом:

Значения ингибирования роста опухоли (ИРО), как показатель активности in vivo, рассчитывали с помощью внутренне статистической программы Roche, как описано выше в примере 12.1.
В конце исследования (сутки 48) мышей умерщвляли, удаляли опухоли, взвешивали и готовили суспензии из одиночных клеток путем ферментативного расщепления коллагеназой V и ДНКазой для последующего анализа FACS. Одиночные клетки окрашивали в отношении CD45 и CD3 человека и анализировали на FACS BDFortessa.
На Фиг. 32 показана кинетика роста опухолей (среднее, + СПС) для всех групп обработки, соответствующие значения ИРО для каждой группы обработки приведены в таблице 35 ниже. Как описано в данном документе, монотерапия СЕА-ТСВ индуцировала ингибирование роста опухолей со значением ИРО 84%. При этом комбинированная обработка CEA-CD28 (SA_вариант 8) приводила к превосходящему ингибированию роста опухолей (ИРО: 101%). Кроме того, данные иммуно-ФД (Фиг. 3) по опухолям от животных, умерщвленных на момент завершения исследования, показали, что сильное ингибирование роста опухолей, индуцированное CEA-CD28 (SA_вариант 8) в комбинации с СЕА-ТСВ, также отображается в повышении частоты Т-клеток внутри опухоли. На Фиг. 33А приведены репрезентативные точечные графики окрашенных суспензий одиночных опухолевых клеток для каждой группы обработки. Обобщенные данные по инфильтрации CD3+ Т-клеток приведены на Фиг. 33Б.

Пример 14
Создание и выработка биспецифических антигенсвязывающих молекул, нацеленных на CD28 и молекулу адгезии эпителиальных клеток (ЕрСАМ), HER3, CD30 или гликопротеин трофобластов (TPBG)
14.1 Клонирование биспецифических антигенсвязывающих молекул, нацеленных на CD28 и ЕрСАМ, HER3, CD30 или TPBG
Для создания экспрессионных плазмид использовали последовательности соответствующих вариабельных доменов, которые субклонировали в рамке с соответствующими константными областями, которые были предварительно вставлены в соответствующий реципиентный экспрессионный вектор млекопитающих. В Fc-домене мутации Pro329Gly, Leu234Ala и Leu235Ala (PG-LALA) были внесены в константную область тяжелых цепей IgG1 человека для устранения связывания с Fc-гамма-рецепторами в соответствии с методом, описанным в публикации заявки на Международный патент № WO 2012/130831. Для создания биспецифических антител Fc-фрагменты содержали мутации типа «выступ» (мутации S354C/T366W, нумерация в соответствии с индексом EU по Kabat) или типа «впадина» (мутации Y349C/T366S/L368A/Y407V, нумерация в соответствии с индексом EU по Kabat) для предотвращения неправильного спаривания тяжелых цепей. Чтобы предотвратить неправильное спаривание легких цепей в биспецифических антигенсвязывающих молекулах, в один связывающий фрагмент вносили обмен доменов VH/VL или СН1/С-каппа (технология CrossFab). В другом связывающем фрагменте в домены СН1 и С-каппа вносили заряды, как описано в публикации заявки на Международный патент № WO 2015/150447.
Создание и подготовка анти-ЕрСАМ антитела МТ201 (адекатумумаб) описаны в патенте США №7632925 В2. Получение анти-HER3 антитела (лумретузумаб) описано в WO 2011/076683 A1. Анти-CD30 антитело брентуксимаб описано в WO 02/34661 А2. Создание и подготовка анти-TPBG антител, например, FAB091, описаны в WO 2017/072207 А1.
Клонировали следующие молекулы, схематические иллюстрации которых показаны на Фиг. 34А-34Г:
Молекула 14А: ЕрСАМ (MT201)-CD28 (SA_вариант 15) формата 1+1, биспецифическая молекула huIgG1 PG-LALA CrossFab с обменом VH/VL в Fab-фрагменте (выступ) CD28(SA вариант 15) и заряженными модификациями в Fab-фрагменте (впадина) ЕрСАМ(МТ201) (Фиг. 34А), содержащая аминокислотные последовательности SEQ ID NO: 366, 367, 390 и 391 (Р1АЕ9051).
Молекула 14 В: HER3 (лумретузумаб)-CD28 (SA_вариант 15) формата 1+1, биспецифическая молекула huIgG1 PG-LALA кросс-Fab с заряженными модификациями в Fab-фрагменте (выступ) CD28(SA вариант 15) и обменом VH/VL в анти-НЕИЗ Fab-фрагменте (впадина) (Фиг. 34Б), содержащая аминокислотные последовательности SEQ ID NO: 357, 358, 392 и 393 (P1AF0151).
Молекула 14С: СЕА (лумретузумаб)-CD28 (SA_вариант 15) формата 1+1, биспецифическая молекула huIgG1 PG-LALA кросс-Fab с заряженными модификациями в Fab-фрагменте (выступ) CD28(SA_вариант 15) и обменом VH/VL в анти-CD30 Fab-фрагменте (впадина) CEA(CD30) (Фиг. 34В), содержащая аминокислотные последовательности SEQ ID NO: 357, 358, 394 и 395 (P1AF1751).
Молекула 14D: TPBG(FAB091)-CD28 (SA_вариант 15) формата 1+1, биспецифическая молекула huIgG1 PG-LALA кросс-Fab с заряженными модификациями в Fab-фрагменте (выступ) CD28(SA вариант 15) и обменом VH/VL в анти-TPBG Fab-фрагменте (впадина) (Фиг. 34Г), содержащая аминокислотные последовательности SEQ ID NO: 357, 358, 396 и 397 (P1AF1752).
14.2 Выработка молекул
Экспрессией вышеуказанных молекул управляет химерный промотор MPSV или промотор CMV. Полиаденилирование обусловлено синтетической последовательностью сигнала поли-А, расположенной в 3' конце КП. Кроме того, каждый вектор содержит последовательность EBV OriP для аутосомальной репликации.
Антитела и биспецифические антитела создавали с помощью временной трансфекции клеток HEK293 EBNA или клеток СНО EBNA. Клетки центрифугировали и заменяли среду предварительно нагретой средой CD СНО (Thermo Fisher, кат. №10743029). Экспрессионные векторы смешивали в среде CD СНО, добавляли ПЭИ (полиэтиленимин, Polysciences, Inc, кат. №23966-1), перемешивали раствор на вортексе и инкубировали 10 минут при комнатной температуре. После этого клетки (2 млн/мл) смешивали с раствором вектор/ПЭИ, переносили в колбу и инкубировали 3 часа при 37°С во встряхивателе-инкубаторе с атмосферой 5% СО2. После инкубации добавляли среду Excell с добавками (80% от общего объема) (W. Zhou and A. Kantardjieff, Mammalian Cell Cultures for Biologies Manufacturing, DOI: 10.1007/978-3-642-54050-9; 2014). Через сутки после трансфекции добавляли добавки (Feed, 12% от общего объема). Клеточные супернатанты собирали через 7 суток путем центрифугирования и последующей фильтрации (0,2 мкм фильтр) и очищали белки из собранного супернатанта стандартными методами, как указано ниже.
14.3 Очистка молекул
Белки очищали из профильтрованных клеточных культуральных супернатантов согласно стандартным протоколам. Вкратце, Fc-содержащие белки очищали из клеточных культуральных супернатантов с помощью аффинной хроматографии с протеином А (уравновешивающий буфер: 20 мМ цитрат натрия, 20 мМ фосфат натрия, рН 7,5; элюирующий буфер: 20 мМ цитрат натрия, рН 3,0). Элюирование проводили при рН 3,0 с последующей незамедлительной нейтрализацией рН образца. Белок концентрировали путем центрифугирования (Millipore Amicon® ULTRA-15 (Изд. №: UFC 903096) и отделяли агрегированный белок от мономерного белка с помощью эксклюзионной хроматографии в 20 мМ гистидине, 140 мМ хлориде натрия, рН 6,0.
14.4 Аналитические данные для биспецифических CD28-антигенсвязывающих молекул
Концентрации очищенных белков определяли путем измерения поглощения на 280 нм, используя массовый коэффициент экстинкции, рассчитанный на основании аминокислотной последовательности, в соответствии с Расе, et al., Protein Science, 1995, 4, 2411-1423. Чистоту и молекулярную массу белков анализировали с помощью КЭ-ДСН в присутствии или отсутствие восстанавливающего агента, используя LabChipGXII (Perkin Elmer). Определение содержания агрегатов проводили с помощью ВЭЖХ хроматографии при 25°С, используя аналитическую эксклюзионную колонку (TSKgel G3000 SW XL или UP-SW3000), уравновешенную в подвижном буфере (25 мМ K2HPO4, 125 мМ NaCl, 200 мМ L-аргинина моногидрохлорида, рН 6,7, или 200 мМ KH2PO4, 250 мМ КС1, рН 6,2, соответственно). Обобщенные данные по параметрам очистки всех молекул приведены в таблице 36.
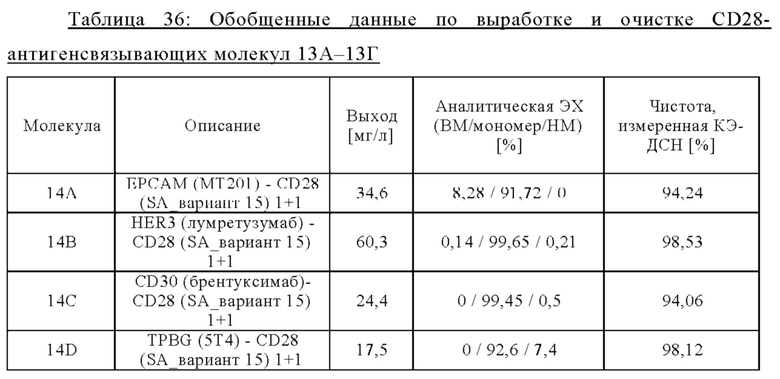
Пример 15
In vitro функциональные характеристики биспецифических антигенсвязывающих молекул, нацеленных на CD28 и ЕрСАМ, HER3, CD30 или TPBG
15.1 Связывание EpCAM-CD28 с клетками, экспрессирующими ЕрСАМ и CD28
Связывание EpCAM-CD28 (молекула 14А), несущей вариант 15 клона CD28 со средней аффинностью (Р1АЕ9051), с ЕрСАМ исследовали, используя клетки НТ-29 (АТСС № НТВ-38), а связывание с CD28 человека исследовали с клетками СНО, экспрессирующими CD28 человека (родительская линия клеток CHO-k1 АТСС № CCL-61, модифицированная для стабильной сверхэкспрессии CD28 человека).
Чтобы оценить связывание, клетки собирали, подсчитывали, проверяли на жизнеспособность и ресуспендировали при 0,5 млн клеток/мл в буфере FACS (eBioscience, кат. №00-4222-26). 5Е4 клеток инкубировали в круглодонных 96-луночных планшетах в течение 1 ч при 4°С с возрастающими концентрациями конструкции EpCAM-CD28 (10 пМ - 500 нМ). Потом клетки дважды промывали холодным буфером FACS, инкубировали еще 30 мин при 4°С с РЕ-конъюгированным, козьим античеловеческим РЕ (Jackson ImmunoReserach, кат. №109-116-098), дважды промывали холодным буфером FACS, центрифугировали и ресуспендировали в 85 мкл буфера FACS с ДАФИ (Roche, кат. №10236276001), разведенным 1:10000. Для отслеживания неспецифических взаимодействий связывания между конструкциями и клетками в качестве отрицательного контроля был включен анти-DP47 IgG. Связывание оценивали методом проточной цитометрии с помощью FACS Fortessa (BD, программное обеспечение FACS Diva). Кривые связывания получали, используя GraphPadPrism7.
In vitro анализ связывания клеток подтвердил, что биспецифическое агонистическое антитело EpCAM-CD28 (Р1АЕ9051) связывается с CD28 человека (Фиг. 35А), а также с ЕрСАМ человека на клетках НТ-29 (Фиг. 35Б) зависимым от концентрации образом. Как и ожидалось, с анти-DP47 IgG связывание обнаружено не было, что свидетельствует о том, что обнаружение связывания обусловлено специфическим связыванием CD28 и ЕрСАМ соответствующими нацеленными фрагментами.
15.2 In vitro функциональные характеристики молекулы ЕрСАМ-CD28 на основании репортерного анализа с IL-2
Чтобы оценить способность EpCAM-CD28 (молекула 14А) поддерживать анти-CD3-опосредованную активацию Т-клеток, репортерные клетки IL-2 (Promega, кат. №J1651) служили в качестве эффекторных клеток (линия Т-клеток Jurkat, которая экспрессирует репортер люциферазы под управлением промотора IL-2), а НТ-29 служили в качестве опухолевых мишеней. 1Е4 опухолевых клеток-мишеней инкубировали в белых плоскодонных 96-луночных планшетах в течение 6 ч при 37°С с 5Е4 репортерных клеток IL-2 (Э:М 5:1) в присутствии 10 нМ анти-CD3 (eBioscience №16-0037-85), одного или в комбинации с возрастающими концентрациями конструкции EpCAM-CD28 (24 пМ 100 нМ). Перед измерением планшеты инкубировали при комнатной температуре в течение 15 мин, а потом к клеткам добавляли 100 μл субстрата (раствор ONE-Glo, Promega, кат. № Е6120). Через 10 мин инкубации при комнатной температуре в темноте измеряли люминесценцию (импульсов/с) с помощью Tecan Spark 10 М.
Оценивали активацию Т-клеток в комбинации с постоянным субоптимальным стимулом анти-CD3. С этой целью репортерные IL-2 клетки Jurkat культивировали вместе с экспрессирующими ЕрСАМ клетками НТ29 в течение 6 ч в присутствии возрастающих концентраций EpCAM-CD28 (Р1АЕ9051) и фиксированной ограничивающей концентрацией анти-CD3 клона IgG ОКТЗ (10 нМ). Как изображено на Фиг. 35С, EpCAM-CD28 был способен усиливать активацию Т-клеток, на что указывает повышенная выработки IL-2 в Т-клетках, подвергнутых субоптимальной стимуляции CD3, зависимым от концентрации образом.
15.3 Связывание HER3-CD28 с клетками, экспрессирующими HER3 и CD28
Связывание HER3-CD28 (молекула 14В), несущей вариант 15 клона CD28 со средней аффинностью (P1AF0151), с HER3 исследовали, используя клетки Т-47D (АТСС № НТВ-133), а связывание с CD28 человека исследовали с клетками СНО, экспрессирующими CD28 человека (родительская линия клеток CHO-k1 АТСС № CCL-61, модифицированная для стабильной сверхэкспрессии CD28 человека).
Чтобы оценить связывание, клетки собирали, подсчитывали, проверяли на жизнеспособность и ресуспендировали при 0,5 млн клеток/мл в буфере FACS (eBioscience, кат. №00-4222-26). 5Е4 клеток инкубировали в круглодонных 96-луночных планшетах в течение 1 ч при 4°С с возрастающими концентрациями конструкции HER3-CD28 (10 пМ - 500 нМ). Потом клетки дважды промывали холодным буфером FACS, инкубировали еще 30 мин при 4°С с РЕ-конъюгированным, козьим античеловеческим РЕ (Jackson ImmunoReserach, кат.№109-116-098), дважды промывали холодным буфером FACS, центрифугировали и ресуспендировали в 85 мкл буфера FACS с ДАФН (Roche, кат. №10236276001), разведенным 1:10000. Для отслеживания неспецифических взаимодействий связывания между конструкциями и клетками в качестве отрицательного контроля был включен анти-ОР47 IgG. Связывание оценивали методом проточной цитометрии с помощью FACS Fortessa (BD, программное обеспечение FACS Diva). Кривые связывания получали, используя GraphPadPrism7.
Анализ FACS
Чтобы оценить относительный уровень HER3 на поверхности T-47D, клетки 2Е5 центрифугировали при 480xg в течение 5 мин и промывали ФСБ. Окрашивание поверхности в отношении HER3 (АРС, античеловеческий, BioLegend №324708) проводили в соответствии с указаниями поставщика. Клетки один раз промывали 150 мкл/лунка ФСБ и ресуспендировали в 150 ил/лунка буфера ФСБ и анализировали, используя BD FACS Fortessa.
In vitro анализ связывания клеток подтвердил, что биспецифическое агонистическое антитело HER3-CD28 (молекула 14В) связывается с CD28 человека (Фиг. 36А), а также с HER3 человека (Фиг. 36Б) на клетках зависимым от концентрации образом. Как и ожидалось, с анти-ОР47 IgG связывание обнаружено не было, что свидетельствует о том, что обнаружение связывания обусловлено специфическим связыванием CD28 и HER3 соответствующими нацеленными фрагментами.
15.4 In vitro функциональные характеристики молекулы HER3-CD28 на основании репортерного анализа с IL-2
Чтобы оценить способность HER3-CD28 (молекула 14 В) поддерживать анти-СО3-опосредованную активацию Т-клеток, репортерные клетки IL-2 (Promega, кат. № J1651) служили в качестве эффекторных клеток (линия Т-клеток Jurkat, которая экспрессирует репортер люциферазы под управлением промотора IL-2), a T-47D служили в качестве опухолевых мишеней. 1Е4 опухолевых клеток-мишеней инкубировали в белых плоскодонных 96-луночных планшетах в течение 6 ч при 37°С с 5Е4 репортерных клеток IL-2 (Э:М 5:1) в присутствии 10 нМ анти-CD3 (eBioscience №16-0037-85), одного или в комбинации с возрастающими концентрациями конструкции HER3-CD28 (24 пМ - 100 нМ). Перед измерением планшеты инкубировали при комнатной температуре в течение 15 мин, а потом к клеткам добавляли 100 мкл субстрата (раствор ONE-Glo, Promega, кат. №Е6120). Через 10 мин инкубации при комнатной температуре в темноте измеряли люминесценцию (импульсов/с) с помощью Tecan Spark 10 М.
Оценивали активацию Т-клеток в комбинации с постоянным субоптимальным стимулом анти-CD3. С этой целью репортерные IL-2 клетки Jurkat культивировали вместе с экспрессирующими ЕрСАМ клетками НТ29 в течение 6 ч в присутствии возрастающих концентраций HER3-CD28 (P1AF0151) и фиксированной ограничивающей концентрацией анти-CD3 клона IgG ОКТ3 (10 нМ). Как изображено на Фиг. 36В, EpCAM-CD28 был способен усиливать активацию Т-клеток, т.е. выработку IL-2 в Т-клетках, подвергнутых субоптимальной стимуляции CD3, зависимым от концентрации образом.
15.5 In vitro функциональные характеристики агонистических антител к TPBG-CD28 и CD30-CD28 в анализе МКПК
Способность TPBG(5T4)-CD28 (молекула 14D) и CD30-CD28 (молекула 14С) усиливать активацию Т-клеток, опосредованную анти-CD3 стимуляцией, будут оценивать с Т-клетками первичных МКПК от здоровых доноров в качестве эффекторных клеток и экспрессирующими 5Т4 клетками, такими как JIMT-1, NCI-H1975, NCI-N87 и Calu-1, или экспрессирующими CD30 клетками, такими как KARPAS-299, соответственно. В этом анализе опухолевые клетки-мишени 1Е4 будут инкубировать в круглодонных 96-луночных планшетах в течение 5 суток при 37°С с 1Е5 (Э:М 10:1) в присутствии субоптимальной концентрации анти-CD3 (eBioscience №16-0037-85, клон ОКТ3), одного или в комбинации с возрастающими концентрациями агонистических конструкций CD28 (24 пМ 100 нМ). Функциональность нацеленных на CD28 молекул будут оценивать методом проточной цитометрии, измеряя маркеры активации Т-клеток (CD25, CD69) и пролиферацию Т-клеток (разведение CFSE).
Пример 16
Создание и выработка биспецифических антигенсвязывающих молекул, нацеленных на CD28 и антиген клеточной поверхности множественной миеломы (ММ)
16.1 Клонирование биспецифических антигенсвязывающих молекул, нацеленных на CD28 и антиген клеточной поверхности множественной миеломы (ММ)
Для создания экспрессионных плазмид использовали последовательности соответствующих вариабельных доменов, которые субклонировали в рамке с соответствующими константными областями, которые были предварительно вставлены в соответствующий реципиентный экспрессионный вектор млекопитающих. В Fc-домене мутации Pro329Gly, Leu234Ala и Leu235Ala (PG-LALA) были внесены в константную область тяжелых цепей IgG1 человека для устранения связывания с Fc-гамма-рецепторами в соответствии с методом, описанным в публикации заявки на Международный патент № WO 2012/130831.
Для создания биспецифических антител Fc-фрагменты содержали мутации типа «выступ» (мутации S354C/T366W, нумерация в соответствии с индексом EU по Kabat) или типа «впадина» (мутации Y349C/T366S/L368A/Y407V, нумерация в соответствии с индексом EU по Kabat) для предотвращения неправильного спаривания тяжелых цепей. Чтобы предотвратить неправильное спаривание легких цепей в биспецифических антигенсвязывающих молекулах, в один связывающий фрагмент вносили обмен доменов VH/VL или СН1/С-каппа (технология CrossFab). В другом связывающем фрагменте в домены СН1 и С-каппа вносили заряды, как описано в публикации заявки на Международный патент № WO 2015/150447.
Ahth-GPRC5D антитело 5Е11 представляет собой гуманизированную версию клона 5Е11, описанного в WO 2019/154890 A1. Ahth-CD38 антитела, например, даратумумаб, описаны в WO 2006/99875 А1. Создание и подготовка анти-ВСМА антител описаны в WO 2016/166629 А1.
Клонировали следующие молекулы, схематические иллюстрации которых показаны на Фиг. 37А-37В:
Молекула 16А: GPRC5D (5E11)-CD28 (SA_вариант 15) формата 1+1, биспецифическая молекула huIgG1 PG-LALA CrossFab с обменом VH/VL в Fab-фрагменте (выступ) CD28(SA вариант 15) и заряженными модификациями в Fab-фрагменте (впадина) GPRC5D (5Е11) (Фиг. 37А), содержащая аминокислотные последовательности SEQ ID NO: 366, 367, 398 и 399 (P1AF1272).
Молекула 16В: GPRC5D (5Е11) -CD28 (SA_вариант 8) формата 1+1, биспецифическая молекула huIgG1 PG-LALA CrossFab с заряженной модификацией в Fab-фрагменте (выступ) CD28(SA вариант 15) и обменом VH/VL в Fab-фрагменте (впадина) GPRC5D (5Е11) (Фиг. 37А), содержащая аминокислотные последовательности SEQ ID NO: 364, 365, 398 и 399 (P1AF2954).
Молекула 16С: CD38 (даратумумаб)-CD28 (SA_вариант 15) формата 1+1, биспецифическая молекула huIgG1 PG-LALA кросс-Fab с заряженными модификациями в Fab-фрагменте (выступ) CD28(SA_вариант 15) и обменом VH/VL в анти-С038 Fab-фрагменте (впадина) (Фиг. 37Б), содержащая аминокислотные последовательности SEQ ID NO: 357, 358, 400 и 401 (Р1АЕ9038).
Молекула 16D: анти-ВСМА-С028 (SA_вариант 15) формата 1+1, биспецифическая молекула huIgG1 PG-LALA CrossFab с обменом VH/VL в Fab-фрагменте (выступ) CD28(SA_вариант 15) и заряженными модификациями в анти-ВСМА Fab-фрагменте (впадина) (Фиг. 34А), содержащая аминокислотные последовательности SEQ ID NO: 366, 367, 402 и 403 (Р1АЕ9053).
16.2 Выработка молекул
Экспрессией вышеуказанных молекул управляет химерный промотор MPSV или промотор CMV. Полиаденилирование обусловлено синтетической последовательностью сигнала поли-А, расположенной в 3' конце КП. Кроме того, каждый вектор содержит последовательность EBV OriP для аутосомальной репликации.
Антитела и биспецифические антитела создавали с помощью временной трансфекции клеток HEK293 EBNA или клеток СНО EBNA. Клетки центрифугировали и заменяли среду предварительно нагретой средой CD СНО (Thermo Fisher, кат. №10743029). Экспрессионные векторы смешивали в среде CD СНО, добавляли ПЭИ (полиэтиленимин, Polysciences, Inc, кат. №23966-1), перемешивали раствор на вортексе и инкубировали 10 минут при комнатной температуре. После этого клетки (2 млн/мл) смешивали с раствором вектор/ПЭИ, переносили в колбу и инкубировали 3 часа при 37°С во встряхивателе-инкубаторе с атмосферой 5% СО2. После инкубации добавляли среду Excell с добавками (80% от общего объема) (W. Zhou and A. Kantardjieff, Mammalian Cell Cultures for Biologies Manufacturing, DOI: 10.1007/978-3-642-54050-9; 2014). Через сутки после трансфекции добавляли добавки (Feed, 12% от общего объема). Клеточные супернатанты собирали через 7 суток путем центрифугирования и последующей фильтрации (0,2 мкм фильтр) и очищали белки из собранного супернатанта стандартными методами, как указано ниже.
В альтернативном варианте описанные в данном документе антитела и биспецифические антитела были изготовлены Evitria с использованием их собственной векторной системы с помощью стандартных (не на основе ПНР) методик клонирования и использованием адаптированных к суспензии клеток СНО К1 (изначально полученных от АТСС и адаптированных для бессывороточного роста в суспензионной культуре в Evitria). Для выработки Evitria использовали их собственные не содержащую животных компонентов и не содержащую сыворотку среды (eviGrow и eviMake2) и их собственный трансфекционный реагент (eviFect). Супернатант собирали путем центрифугирования и последующей фильтрации (0,2 мкм фильтр) и очищали белки из собранного супернатанта стандартными методами.
16.3 Очистка молекул
Белки очищали из профильтрованных клеточных культуральных супернатантов согласно стандартным протоколам. Вкратце, Fc-содержащие белки очищали из клеточных культуральных супернатантов с помощью аффинной хроматографии с протеином А (уравновешивающий буфер: 20 мМ цитрат натрия, 20 мМ фосфат натрия, рН 7,5; элюирующий буфер: 20 мМ цитрат натрия, рН 3,0). Элюирование проводили при рН 3,0 с последующей незамедлительной нейтрализацией рН образца. Белок концентрировали путем центрифугирования (Millipore Amicon® ULTRA-15 (Изд. №: UFC 903096) и отделяли агрегированный белок от мономерного белка с помощью эксклюзионной хроматографии в 20 мМ гистидине, 140 мМ хлориде натрия, рН 6,0.
16.4 Аналитические данные для биспецифических CD28-антигенсвязывающих молекул
Концентрации очищенных белков определяли путем измерения поглощения на 280 нм, используя массовый коэффициент экстинкции, рассчитанный на основании аминокислотной последовательности, в соответствии с Расе, et al., Protein Science, 1995, 4, 2411-1423. Чистоту и молекулярную массу белков анализировали с помощью КЭ-ДСН в присутствии или отсутствие восстанавливающего агента, используя LabChipGXII или LabChip GX Touch (Perkin Elmer). Определение содержания агрегатов проводили с помощью ВЭЖХ-хроматографии при 25°С, используя аналитическую эксклюзионную колонку (TSKgel G3000 SW XL или UP-SW3000), уравновешенную в подвижном буфере (25 мМ K2HPO4, 125 мМ NaCl, 200 мМ L-аргинина моногидрохлорида, рН 6,7, или 200 мМ KH2PO4, 250 мМ KCl, рН 6,2, соответственно). Обобщенные данные по параметрам очистки молекул приведены в таблице 37.
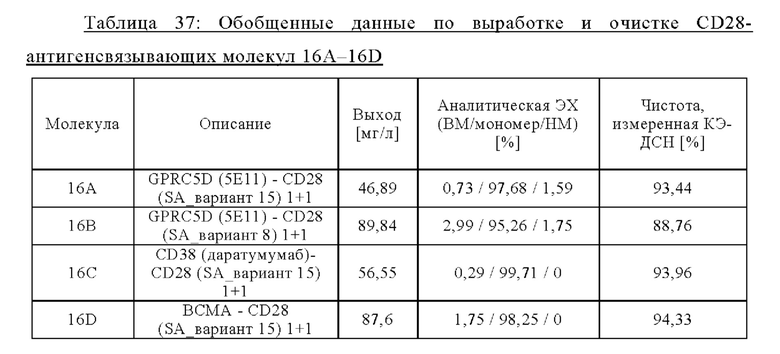
Пример 17
In vitro функциональные характеристики биспецифических антигенсвязывающих молекул, нацеленных на CD28 и антиген клеточной поверхности множественной миеломы (ММ)
17.1 Связывание биспецифических антигенсвязывающих молекул, нацеленных на CD28 и антиген клеточной поверхности множественной миеломы (ММ) с клетками, сверхэкспрессирующими указанную мишень
Чтобы измерить связывание с GPRC5D, ВСМА, CD38 или CD28 мы проводили анализ связывания на основе FACS на указанных линиях клеток множественной миеломы (Lombardi et al., Molecular characterization of human multiple myeloma cell lines by integrative genomics: insights into the biology of the diseas; Genes Chromosomes Cancer. 2007, 46(3), 226-38) или трансфектантах СНО, которые были трансдуцированы для стабильной сверхэкспрессии GPRC5D человека или CD28 человека: связывание с ВСМА оценивали, используя клетки IM-9 (АТСС® CCL-159), связывание с CD38 оценивали, используя клетки OCI-Ly18 (DSMZ АСС 699), связывание с GPRC5D оценивали, используя клетки CHO-hGPRC5D, и связывание с CD28 оценивали, используя клетки CHO-hCD28.
IM-9 и OCI-Ly18 культивировали в соответствии с инструкциями производителя, стабильные трансфектанты СНО (родительская линия клеток CHO-k1 АТСС № CCL-61) культивировали в F-12K, дополненной 10% ФТС. Вкратце, суспензионные клетки собирали, подсчитывали и проверяли на жизнеспособность. Адгезивные клетки СНО отсоединяли, используя буфер для клеточной диссоциации (Gibco), подсчитывали и проверяли на жизнеспособность. Все последующие этапы проводили при 4°С.
Клетки один раз промывали в буфере FACS (ФСБ, 2% фетальная бычья сыворотка; 1% 0,5 М ЭДТА, рН 8; 0,25% NaN3 азида натрия) и ресуспендировали в буфере FACS при 1 млн клеток на мл. Высевали 0,1 млн клеток на лунку круглодонного 96-луночного планшета, еще один раз промывали буфером FACS и сливали супернатанты. Клетки окрашивали в общем объеме 50 мкл на лунку с возрастающими концентрациями указанных биспецифических молекул CD28 (0,07-300 нМ) в течение 30 мин при 4°С. Клетки дважды промывали буфером FACS и инкубировали в течение 30 мин при 4°С в общем объеме 25 мкл на лунку, содержащую предварительно разведенное вторичное антитело (Alexa Fluor 488-AffiniPure Р(ab')2-фрагмент козьего античеловеческого IgG, специфический к Fcγ-фрагменту (Jackson Immunoresearch, 109-546-008, разведенное 1:100 в буфере FACS, соответствующее 109-606-008 РЕ, Jackson Immunoresearch, разведенное 1:100 в буфере FACS, как указано). Клетки дважды промывали и анализировали на проточном цитометре BD Fortessa, оборудованном программным обеспечением FACS Diva. Кривые связывания и значения ЕС50 получали, используя GraphPadPrism6.
На Фиг. 38А-38Е показано, что все биспецифические молекулы CD28 способны связывать CD28 человека (А), а также соответствующую вторую мишень, а именно CD38 человека, ВСМА человека или GPRC5D человека зависимым от концентрации образом. Вкратце, молекула GPRC5D-CD28 имеет наименьшее значение ЕС50 для связывания с CD28 человека (таблица 37), но достигает наименьшего максимального связывания по сравнению с двумя другими биспецифическими молекулами CD28.
Обе нацеленные на CD38 и на ВСМА молекулы CD28 демонстрируют зависимое от концентрации связывание с CD38 и ВСМА, соответственно, и достигают насыщения в оцениваемом диапазоне концентраций (значения ЕС50 для связывания обобщены в таблице 38). В противоположность этому, молекула GPRC5D-CD28 связывается зависимым от концентрации образом, но не достигает насыщения в том же диапазоне концентраций, что указывает на более низкую аффинность GPRC5D по сравнению со связывающими CD38 или ВСМА фрагментами, включенными в эти молекулы.
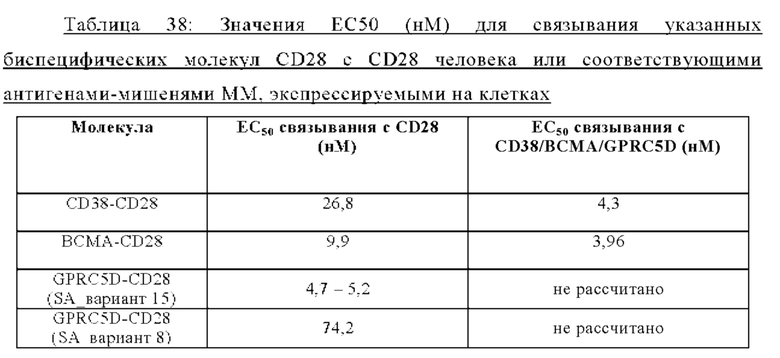
17.2 In vitro функциональные характеристики на основании репортерного анализа с IL-2 (функциональные характеристики активации Т-клеток)
Чтобы оценить способность биспецифических антигенсвязывающих молекул CD38-CD28, BCMA-CD28 и GPRC5D-CD28 поддерживать ТСВ-опосредованную активацию Т-клеток, репортерные клетки IL-2 (Promega, кат. №J1651) служили в качестве эффекторных клеток (линия Т-клеток Jurkat, которая экспрессирует репортер люциферазы под управлением промотора IL-2), а клетки NCI-H929, положительные в отношении CD38, ВСМА, а также GPRC5D, служили в качестве опухолевых мишеней.
Вкратце, 5×103 опухолевых клеток-мишеней инкубировали в белых плоскодонных 384-луночных планшетах (353988 Falcon™, 384-луночный плоскодонный обработанный микропланшет для тканевого культивирования) в течении 5 ч и, соответственно, 22 ч при 37°С с 2,5×104 IL-2 репортерных клеток (Э:М 5:1) в присутствии 1 нМ GPRC5D-TCB, одного или в комбинации с возрастающими концентрациями биспецифических молекул CD28 (12,2 50 нМ). Перед измерением планшеты инкубировали при комнатной температуре в течение 15 мин, а потом к клеткам добавляли 20 мкл субстрата (раствор ONE-Glo, Promega, кат. №Е6120). Через 10 мин инкубации при комнатной температуре в темноте измеряли люминесценцию (импульсов/с) с помощью Tecan Spark 10 М.
Как изображено на Фиг. 39А-39Е, ни одна из нацеленных на ММ молекул CD28 не индуцировали активацию репортерных клеток IL2 Jurkat в отсутствие сигнала ТСВ (яркая серая линия, Фиг. 39А 9Е). Как и ожидалось с этими репортерными клетками IL-2, также отсутствует существенная индукции активации репортерных клеток в присутствии 1 нМ GPRC5D ТСВ, как в ранний момент времени (5 ч), так и в поздний момент времени (через 22 ч).
Однако все нацеленные на ММ молекулы способны индуцировать существенную, независимую от концентрации и времени активацию репортерных клеток IL-2 в присутствии 1 нМ GPRC5D-TCB. В то время как молекулы CD38-CD28 и BCMA-CD28 способны индуцировать активацию репортерных клеток IL2 уже через 5 ч (Фиг. 39А и 39В), GPRC5D-CD28 достигает аналогичных максимальных уровней только в более поздний оцениваемый момент времени (22 ч, Фиг. 39Е), что указывает на разную кинетику активности.
AHTH-GPRC5D/анти-CD3 биспецифическое антитело (GPRC5D ТСВ, Фиг. 37Г), используемое в экспериментах, готовили аналогично с СЕАСАМ5 ТСВ, как описано в примере 8 и в WO 2016/079076 A1. GPRC5D ТСВ содержит аминокислотные последовательности SEQ ID NO: 398, SEQ ID NO: 399, SEQ ID N0:404 и SEQ ID N0:405.
17.3 Опосредованный Т-клетками лизис линии клеток множественной миеломы
Чтобы оценить способность CD38-CD28, BCMA-CD28 и GPRC5D-CD28 усиливать опосредованный GPRC5D ТСВ лизис линии клеток множественной миеломы, 1,5×105 человеческих пан-Т-эффекторных клеток инкубировали с 3×104 клеток-мишеней NCI-H929 в конечном соотношении Э:М 1:1 приблизительно 22 ч. Пан-Т-клетки выделяли из МКПК с помощью MACS, используя набор для выделения пан-Т-клеток (Miltenyi Biotec, кат. №130-096-535) в соответствии с инструкциями производителя. GPRC5D ТСВ добавляли в возрастающих концентрациях (0,064 пМ-1 нМ), разные нацеленные на ММ биспецифические CO28-антигенсвязывающие молекулы добавляли в фиксированной концентрации 0,2 нМ. Лизис опухолевых клеток оценивали следующим образом: Аналитические планшеты центрифугировали в течение 5 минут и переносили по 50 мкл супернатанта на лунку в новый 96-луночный плоскодонный планшет. Для нормализации максимальный лизин клеток-мишеней (=100%) индуцировали путем инкубации клеток-мишеней с конечной концентрацией 1% Тритон Х-100. Минимальный лизис (=0%) относится к клеткам-мишеням, которые инкубировали с эффекторными клетками, но без какой-либо биспецифической конструкции или ТСВ. После инкубации в течение ночи приблизительно 22 ч при 37°С, 5% CO2, измеряли высвобождение ЛДГ апоптотических/некротических клеток-мишеней в супернатант, используя набор для обнаружения ЛДГ (Roche Applied Science, №11 644 793 001), в соответствии с инструкциями производителя.
Как проиллюстрировано на Фиг. 40А-40В, GPRC5D ТСВ индуцирует зависимый от концентрации лизис клеток NCI-H929. Все три оцениваемые нацеленные на ММ биспецифические CD28-антигенсвязывающие молекулы способны значительно усиливать эффективность по сравнению с монотерапией ТСВ при введении в фиксированной концентрации 0,2 нМ. Кроме того, ни одна из нацеленных на MM CD28 биспецифических CD28-антигенсвязывающих молекул не индуцирует лизис опухолевых клеток в отсутствие ТСВ, что поддерживает предположение о зависимости нацеленных на ММ молекул от сигнала ТСВ.
В таблице 39 обобщены значения ЕС50, а также площади под кривыми, полученные из данных, приведенных на Фиг. 40А-40В. Значения ЕС50 рассчитывали, используя GraphPadPrism6.
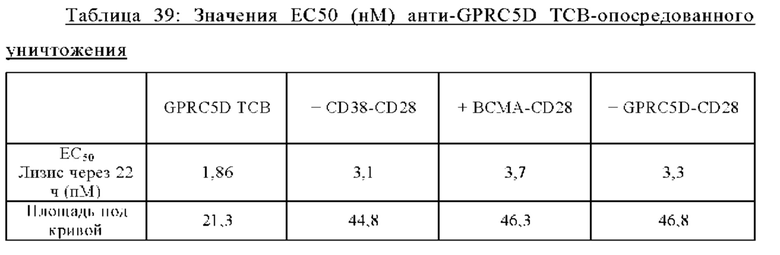
Пример 18
Создание и выработка биспецифических антигенсвязывающих молекул, нацеленных на CD28 и CD19 или CD79b
18.1 Клонирование биспецифических антигенсвязывающих молекул, нацеленных на CD28 и CD19 или CD79b
Создание и подготовка антител к CD19 описаны в WO 2017/55541 А1 или WO 2017/55328 А1. В частности, клон CD19 2В11 описан в WO 2017/55328 А1, которая включена в данный документ посредством ссылки. Клон CD79b huMA79b.v28 (соответствует полатузумабу) в контексте даного документа описан в WO 2009/012268 А. Для создания соответствующих экспрессионных плазмид использовали последовательности соответствующих вариабельных доменов, которые субклонировали в рамке с соответствующими константными областями, которые были предварительно вставлены в соответствующий реципиентный экспрессионный вектор млекопитающих. Схематическое описание полученных в результате молекул приведено на Фиг. 41А и 41Б, соответственно. Мутации Pro329Gly, Leu234Ala и Leu235Ala (PG-LALA) были внесены в константную область тяжелых цепей IgG1 человека для устранения связывания с Fc-гамма-рецепторами. Для создания несимметричных биспецифических антител Fc-фрагменты содержали мутации типа «выступ» или «впадина», чтобы предотвратить неправильное спаривание тяжелых цепей. Чтобы предотвратить неправильное спаривание легких цепей в би- и мультиспецифических конструкциях антител, в один связывающий фрагмент вносили обмен доменов VH/VL или СН1/С-каппа (технология CrossFab). В другом связывающем фрагменте в домены СН1 и С-каппа вносили заряды.
Клонировали следующие молекулы, схематические иллюстрации которых показаны на Фиг. 41А и 41Б:
Молекула 18А: CD19 (8В8-2 В11)-CD28 (SA_v29) 1+1, биспецифическая huIgG1 PG-LALA молекула кросс-Fab с зарядовыми модификациями в Fab (выступ) CD28 v29 и обменом VH/VL в Fab (впадина) CD19 (2В11) (Фиг. 41А). Молекула содержит аминокислотные последовательности тяжелой цепи SEQ ID NO: 118 и 430 и аминокислотные последовательности легкой цепи SEQ ID NO: 65 и 431 (Р1АЕ8002).
Молекула 18 В: CD19 (8В8-2 В11)-CD28 (SA_v15) 1+1, биспецифическая huIgG1 PG-LALA молекула кросс-Fab с зарядовыми модификациями в Fab (выступ) CD28 v15 и обменом VH/VL в Fab (впадина) CD19 (2В11) (Фиг. 41А). Молекула содержит аминокислотные последовательности тяжелой цепи SEQ ID NO: 116 и 430 и аминокислотные последовательности легкой цепи SEQ ID NO: 121 и 431 (Р1АЕ9040).
Молекула 18С: CD19 (8В8-2В11)-CD28 (SA_v8) 1+1, биспецифическая huIgG1 PG-LALA молекула кросс-Fab с зарядовыми модификациями в Fab (выступ) CD28 v8 и обменом VH/VL в Fab (впадина) CD19 (2В11) (Фиг. 41А). Молекула содержит аминокислотные последовательности тяжелой цепи SEQ ID NO: 114 и 430 и аминокислотные последовательности легкой цепи SEQ ID NO: 122 и 431 (P1AF0175).
Молекула D: CD19 (8В8-2В11)-CD28 (SA_v11) 1+1, биспецифическая huIgG1 PG-LALA молекула кросс-Fab с зарядовыми модификациями в Fab (выступ) CD28 v11 и обменом VH/VL в Fab (впадина) CD19 (2В11) (Фиг. 2Б). Молекула содержит аминокислотные последовательности тяжелой цепи SEQ ID NO: 114 и 430 и аминокислотные последовательности легкой цепи SEQ ID NO: 65 и 431 (P1AF0377).
Молекула Е: CD19 (8В8-2В11)-CD28 (SA_v27) 1+1, биспецифическая huIgG1 PG-LALA молекула кросс-Fab с зарядовыми модификациями в Fab (выступ) CD28 v27 и обменом VH/VL в Fab (впадина) CD19 (2В11) (Фиг. 2Б). Молекула содержит аминокислотные последовательности тяжелой цепи SEQ ID NO: 118 и 430 и аминокислотные последовательности легкой цепи SEQ ID NO: 123 и 431 (P1AF0378).
Молекула F: CD79b (huMA79b.v28)-CD28 (SA_v15) 1+1, биспецифическая huIgG1 PG-LALA молекула кросс-Fab с зарядовыми модификациями в Fab (выступ) CD28 v15 и обменом VH/VL в Fab (впадина) CD79b (huMA79b.v28) (Фиг. 2В). Молекула содержит аминокислотные последовательности тяжелой цепи SEQ ID NO: 116 и 432 и аминокислотные последовательности легкой цепи SEQ ID NO: 121 и 433 (Р1АЕ9039).
18.2 Выработка биспецифических антигенсвязывающих молекул, нацеленных на CD28 и CD19 или CD79b
Экспрессией вышеуказанных молекул управляет промотор CMV. Полиаденилирование обусловлено синтетической последовательностью сигнала поли-А, расположенной в 3' конце КП. Кроме того, каждый вектор содержит последовательность EBV OriP для аутосомальной репликации.
Для выработки всех конструкций клетки HEK293-EBNA, которые росли в суспензии, временно котрансфицировали соответствующими экспрессионными векторами, используя в качестве трансфекционного реагента полиэтиленимин. Клетки центрифугировали и заменяли среду предварительно нагретой средой CD СНО. Экспрессионные векторы смешивали в среде CD СНО, добавляли ПЭИ, перемешивали раствор на вортексе и инкубировали 10 минут при комнатной температуре. После этого клетки смешивали с раствором ДНК/ПЭИ, переносили во встряхиваемую колбу и инкубировали 3 часа при 37°С в инкубаторе с атмосферой 5% CO2. После инкубации добавляли среду Excell с добавками (культуры клеток млекопитающих для производства биопрепаратов, редакторы: Weichang Zhou, Anne Kantardjieff). Через одни сутки после трансфекции добавляли добавки (Feed) (культуры клеток млекопитающих для производства биопрепаратов, редакторы: Weichang Zhou, Anne Kantardjieff). Клеточные супернатанты собирали через 7 суток путем центрифугирования и последующей фильтрации (0,2 мкм фильтр) и очищали стандартными методами.
18.3 Очистка биспецифических антигенсвязывающих молекул, нацеленных на CD28 и CD19 или CD79b
Белки очищали из профильтрованных клеточных культуральных супернатантов согласно стандартным протоколам. Вкратце, Fc-содержащие белки очищали из клеточных культуральных супернатантов с помощью аффинной хроматографии с протеином А (уравновешивающий буфер: 20 мМ цитрат натрия, 20 мМ фосфат натрия, рН 7,5; элюирующий буфер: 20 мМ цитрат натрия, рН 3,0). Элюирование проводили при рН 3,0 с последующей незамедлительной нейтрализацией рН образца. Белок концентрировали путем центрифугирования (Millipore Amicon® ULTRA-15 (Изд. №: UFC903096) и отделяли агрегированный белок от мономерного белка с помощью эксклюзионной хроматографии в 20 мМ гистидине, 140 мМ хлориде натрия, рН 6,0.
18.4 Аналитические данные для биспецифических или триспецифических антител, нацеленных на CD28 и CD19 или CD79b
Концентрацию белка очищенных конструкций определяли путем измерения оптической плотности (ОП) на 280 нм, используя массовый коэффициент экстинкции, рассчитанный на основании аминокислотной последовательности, в соответствии с Расе, et al., Protein Science, 1995, 4, 2411-1423. Чистоту и молекулярную массу белков анализировали с помощью КЭ-ДСН в присутствии или отсутствие восстанавливающего агента, используя LabChipGXII (Perkin Elmer). Определение содержания агрегатов проводили с помощью ВЭЖХ-хроматографии при 25°С, используя аналитическую эксклюзионную колонку (TSKgel G3000 SW XL или UP-SW3000), уравновешенную в подвижном буфере (25 мМ K2HPO4, 125 мМ NaCl, 200 мМ L-аргинина моногидрохлорида, рН 6,7, или 200 мМ KH2PO4, 250 мМ KCl, рН 6,2,соответственно). Обобщенные данные по параметрам очистки всех молекул приведены в таблице 40.

Пример 19
Связывание и кинетический анализ биспецифических антигенсвязывающих молекул, нацеленных на CD28 и CD19 или CD79b
19.1 Кинетический анализ биспецифических антигенсвязывающих молекул, нацеленных на CD79b
Аффинность (KD) huMA79b.v28 к рекомбинантному CD79b-His (Sinobiological №29750-Н08Н) измеряли методом поверхностного плазмонного резонанса или ППР, используя прибор Biacore Т200. Для захвата CD79b-His проводили сопряжение анти-пента-HIS антитела с проточной ячейкой чипа СМ5 с помощью реакции сопряжения аминов. Использовали уровни иммобилизации прибл. 5000 единиц. Потом CD79b-His разводили до концентрации 1 нМ и проводили захват с помощью анти-пента-HIS антитела в течение 10 с при скорости потока 10 μл/мин.
Для определения аффинности (KD) очищенной молекулы F (CD79b (huMA79b.v28)-CD28 (SA вариант 15) 1+1) двукратные серийные разведения очищенной антигенсвязывающей молекулы (диапазон переменных концентраций от 125 до 0,49 нМ) вводили при 30 мкл/мин со временем ассоциации 180 с и временем диссоциации 400 с. Буфер HBS-EP+ (стандартный буфер GE-Healthcare BR-1006-69 в разведении 1:10) вводили для сравнения. Восстановление проводили 10 мМ глицина, рН 2,1 в течение 2x60 с. Константы скорости ассоциации (kon) и константы скорости диссоциации (koff) рассчитывали, используя простую один-к-одному модель связывания Лэнгмюра, путем одновременной аппроксимации сенсограмм ассоциации и диссоциации (Фиг. 42). Равновесную константу диссоциации (KD) рассчитывали как соотношение koff/k0(Kd). Результаты приведены в Таблице 41. Аффинность молекулы F (CD79b (huMA79b.v28)-CD28 (8А_вариант 15) 1+1) к CD79b-His составляет 76 нМ.

19.2 Кинетический анализ биспецифических антигенсвязывающих молекул, нацеленных на CD28
Аффинность (KD) полученных биспецифических антигенсвязывающих молекул к CD28 измеряли методом ППР, используя инструмент ProteOn XPR36 (Biorad) при 25°С с биотинилированным антигеном huCD28-Fc, иммобилизованным на чипах NLC путем захвата нейтравидина. Иммобилизация рекомбинантных антигенов (лиганд): Антиген разводили ФСБТ (10 мМ фосфата, 150 мМ хлорида натрия, рН 7,4, 0,005% Твин 20) до 10 мкг/мл, потом вводили при 30 мкл/минут с переменным временем контакта для обеспечения уровней иммобилизации около 200, 400 или 800 единиц ответа (ЕО) в вертикальной ориентации. Введение аналитов: Для одноразовых измерений кинетики направление введения меняли на горизонтальную ориентацию, двукратные серийные разведения очищенных биспецифических нацеленных на CD19 анти-CD28 вариантов по аффинности (диапазон переменной концентрации от 50 до 3,125 нМ) вводили одновременно при 50 мкл/мин вдоль отдельных каналов 15, со временем ассоциации 150 с и временем диссоциации 450 с. Буфер (ФСБТ) вводили вдоль шестого канала для обеспечения «параллельной» холостой пробы для сравнения. Константы скорости ассоциации (kon) и константы скорости диссоциации (koff) рассчитывали, используя простую один-к-одному модель связывания Лэнгмюра в программном обеспечении ProteOn Manager v3.1, путем одновременной аппроксимации сенсограмм ассоциации и диссоциации. Равновесную константу диссоциации (KD) рассчитывали как соотношение koff/kon. Анализируемые клоны демонстрировали значения KD в широком диапазоне (от 1 до 50 нМ).
Пример 20
Связывание биспецифических агонистических CD28-антигенсвязывающих молекул с экспрессирующими CD28 и CD19 или CD79b клетками
Связывание с CD28 человека исследовали с клетками СНО, экспрессирующими CD28 человека (родительская линия клеток CHO-k1 АТСС №CCL-61, модифицированная для стабильной сверхэкспрессии CD28 человека). Чтобы оценить связывание, клетки собирали, подсчитывали, проверяли на жизнеспособность и ресуспендировали при 2,5×105/мл в буфере FACS (eBioscience, кат. №00-4222-26). 5×104 клеток инкубировали в круглодонных 96-луночных планшетах в течение 2 ч при 4°С с возрастающими концентрациями, связывающих CD28 молекул (1 пМ-100 нМ). Потом клетки три раза промывали холодным буфером FACS, инкубировали еще 60 мин при 4°С с РЕ-конъюгированным, козьим античеловеческим РЕ (Jackson ImmunoReserach, кат. №109-116-098), промывали один раз холодным буфером FACS, центрифугировали и ресуспендировали в 100 мкл буфера FACS. Для отслеживания неспецифических взаимодействий связывания между конструкциями и клетками в качестве отрицательного контроля был включен анти-DP47 IgG. Связывание оценивали методом проточной цитометрии с помощью FACS Fortessa (BD, программное обеспечение FACS Diva). Кривые связывания получали, используя GraphPadPrism6.
Одновалентные IgG-подобные CD28 вариантные конструкции с одним плечом демонстрировали разницу в связывании, как можно видеть на Фиг. 4А-4В.
Связывание CD19 и CD79b исследовали, используя линии В-клеток, экспрессирующие разные уровни CD19 и CD79b: Nalm6 (DSMZ №АСС 128), RCK8 (DSMZ №АСС 561), WSU DLCL2 (DSMZ №АСС 575) и Z138 (подарок от М. Dyer, Univ. of Leicester).
Чтобы оценить связывание, клетки собирали, подсчитывали, проверяли на жизнеспособность и ресуспендировали при 0,5 млн клеток/мл в буфере FACS (eBioscience, кат. №00-4222-26). 5Е4 клеток инкубировали в круглодонных 96-луночных планшетах в течение 1 ч при 4°С с возрастающими концентрациями конструкций CD19-CD28 (или CD79b-CD28) (10 нМ-500 нМ). Потом клетки дважды промывали холодным буфером FACS, инкубировали еще 30 мин при 4°С с РЕ-конъюгированным, козьим античеловеческим РЕ (Jackson ImmunoReserach, кат. №109-116-098), дважды промывали холодным буфером FACS, центрифугировали и ресуспендировали в 85 мкл буфера FACS с ДАФИ (Roche, кат. №10236276001), разведенным 1:10000. Для отслеживания неспецифических взаимодействий связывания между конструкциями и клетками в качестве отрицательного контроля был включен анти-DP47 IgG. Связывание оценивали методом проточной цитометрии с помощью FACS Fortessa (BD, программное обеспечение FACS Diva). Кривые связывания получали, используя GraphPadPrism7. Сравнение связывания CD19-CD28 v15 с разными линиями В-клеток показано на Фиг. 43А и 43Б.
Анализ FACS
Чтобы оценить относительный уровень CD19 на поверхности линий В-клеток (Nalm6, RCK8, WSU DLCL2 и Z138), клетки Fc-блокировали перед окрашиванием, используя блокатор Fc человека (BD, кат. №564220), потом 2×105 клеток центрифугировали при 480×g в течение 5 мин и промывали ФСБ. Окрашивание поверхности в отношении CD19 (BV650, античеловеческий, BioLegend №302238) проводили в соответствии с указаниями поставщика. Клетки один раз промывали 150 μл/лунка ФСБ и ресуспендировали в 150 μл/лунка буфера ФСБ и анализировали, используя BD FACS Fortessa.
In vitro анализ связывания клеток подтвердил, что все агонистические антитела к CD19-CD28 связываются с CD19 человека, а также CD28 человека на клетках зависимым от концентрации образом (Фиг. 44А и 44Б). Как и ожидалось, с анти-DP47 IgG связывание обнаружено не было, что свидетельствует о том, что обнаружение связывания обусловлено специфическим связыванием CD28 и CD19 соответствующими нацеленными фрагментами. Значения ЕС50 для связывания CD28 приведены в таблице 42, а значения ЕС50 для связывания с CD19 приведены в таблице 43.
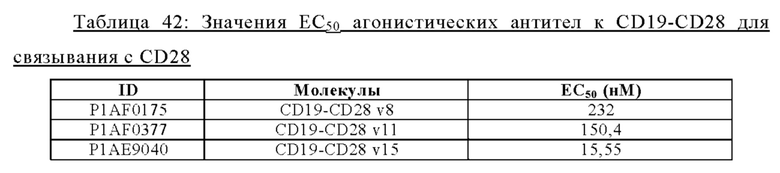

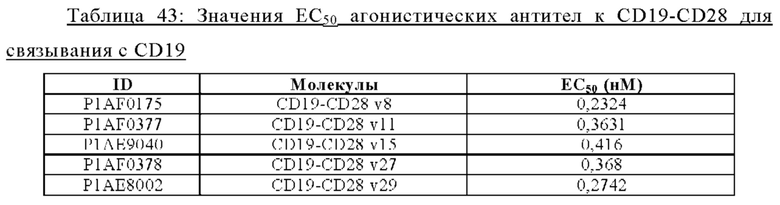
Пример 21
In vitro функциональные характеристики биспецифических агонистических CD28-антигенсвязывающих молекул, нацеленных на CD19 или CD79b
Проводили несколько клеточных in vitro анализов с первичными человеческими МКПК, чтобы оценить активность CD28(SA) и биспецифических нацеленных на CD19 или CD79b CD28-антигенсвязывающих молекул в присутствии или отсутствие сигналов ТКР, обеспечиваемых Т-клеточными биспецифическими (ТСВ) антителами. Пролиферацию Т-клеток, секрецию цитокинов и уничтожение опухолевых клеток, определяемые с помощью проточной цитометрии, ELISA цитокинов и визуализации живых клеток, получали как результаты измерений.
Выделение МКПК
Мононуклеарные клетки периферической крови (МКПК) получали с помощью центрифугирования в градиенте плотности из обогащенных лимфоцитарных препаратов гепаринизированной крови, полученных из лейкоцитаной пленки (Blutspende Zurich). 25 мл крови (разведенной 1:2 в ФСБ) наносили слоем на 15 мл реагента lymphoprep (STEMCELL technologies, кат. №07851) и центрифугировали при комнатной температуре в течение 25 мин при 845×g без перерыва. Содержащую МКПК межфазную поверхность собирали в 50 мл пробирки с помощью 10 мл пипетки. Клетки промывали ФСБ и центрифугировали 5 мин при 611×g. Супернатант сливали, осадок ресуспендировали в 50 мл ФСБ и центрифугировали 5 мин при 304×g. Этап промывки повторяли и центрифугировали при 171×g. Клетки ресуспендировали в RPMI 1640 Glutamax (содержащем 5% человеческую сыворотку, пируват натрия, NEAA, 50 μM 2-меркаптоэтанол, пенициллин/стрептомицин) и обрабатывали для дальнейшего функционального анализа согласно протоколу соответствующего анализа.
In vitro функциональные характеристики молекул CD19-CD28 и CD79b-CD28 на основании репортерного анализа с IL-2
Чтобы оценить способность CD19-CD28 и CD79b-CD28 поддерживать ТСВ-опосредованную активацию Т-клеток, репортерные клетки IL-2 (Promega, кат. №Л651) служили в качестве эффекторных клеток (линия Т-клеток Jurkat, которая экспрессирует репортер люциферазы под управлением промотора IL-2), a Nalm6, RCK8, WSU DLCL2 и Z138 служили в качестве опухолевых мишеней. 2×104 опухолевых клеток-мишеней инкубировали в белых плоскодонных 96-луночных планшетах в течение 6 ч при 37°С с 105 репортерных клеток IL-2 (Э:М 5:1) в присутствии субоптимальных концентраций CD20-TCB (P1AD4071) (10 нМ для Nalm6 или 0,05 нМ для RCK8, WSU DLCL2 и Z138), одного или в комбинации с возрастающими концентрациями конструкций CD19-CD28 (или CD79b-CD28) (0,2 пМ-10 нМ). Перед измерением планшеты инкубировали при комнатной температуре в течение 15 мин, а потом к клеткам добавляли 100 мкл субстрата (раствор ONE-Glo, Promega, кат. №Е6120). Через 10 мин инкубации при комнатной температуре в темноте измеряли люминесценцию (импульсов/с) с помощью Tecan Spark 10М.
In vitro функциональные характеристики CD19-CD28 на основании активации выделенных из МКПК Т-клеток
Чтобы оценить способность CD19-CD28 поддерживать ТСВ-опосредованную активацию Т-клеток, пан-Т-клетки использовали в качестве эффекторных клеток и выделяли из МКПК с помощью MACS, используя набор для выделения пан-Т-клеток (Miltenyi Biotec, кат. №130-096-535) в соответствии с инструкциями производителя, a Nalm6, RCK8, WSU DLCL2 и Z138 служили в качестве опухолевых мишеней. 2×104 опухолевых клеток-мишеней инкубировали в плоскодонных 96-луночных планшетах в течение 48 ч при 37°С с 105 пан-Т-клеток (Э:М 5:1) в присутствии субоптимальных концентраций CD20-TCB (P1AD4071) (10 нМ для Nalm6 или 0,05 нМ для RCK8, WSU DLCL2 и Z138), одного или в комбинации с возрастающими концентрациями конструкций CD19-CD28 (0,2 пМ 10 нМ). Активацию Т-клеток оценивали методом проточной цитометрии. Вкратце, клетки центрифугировали при 480×g в течение 5 мин и промывали ФСБ. Поверхностное окрашивание в отношении CD8 (BV421, античеловеческое, BioLegend №301036), CD4 (РЕ-Су7, античеловеческое, BioLegend №344611), CD25 (BV605, античеловеческое, BioLegend №302632), CD69 (РЕ, античеловеческое, BioLegend №310906) проводили в соответствии с указаниями поставщика. Клетки один раз промывали 150 мкл/лунка ФСБ и ресуспендировали в 150 мкл/лунка буфера ФСБ и анализировали, используя BD FACS Fortessa.
Оценка высвобождения цитокинов
Чтобы оценить способность CD19-CD28 инициировать высвобождение цитокинов в присутствии сигнализации ТКР, 5×105 МКПК инкубировали в 96-луночных планшетах с U-образным дном в течение 48 ч при 37°С в присутствии CD19-CD28 (1 нМ) и субоптимальной концентрации CD20-TCB (P1AD4071) (0,4 пМ). Чтобы оценить способность CD19-CD28 инициировать высвобождение цитокинов в отсутствие сигнализации ТКР, 5×10 МКПК инкубировали в 96-луночных планшетах с U-образным дном в течение 48 ч при 37°С в присутствии возрастающих концентраций конструкций CD19-CD28 (0,08 нМ 100 нМ). Высвобождение цитокинов оценивали с помощью многоканального анализа, проводили скрининг 50 μл/лунка супернатанта в отношении секреции G-CSF, GM-CSF, IFN-γ, IL-1β, IL-2, IL-4, IL-5, IL-6, IL-7, IL-8, IL-10, IL-12 (р70), IL-13, IL-17, МСР-1 (MCAF), MIP-1β и TNF-α, используя 17-канальный анализ цитокинов человека Bio-Plex Pro (Bio-rad, кат. №m5000031yv) в соответствии с указаниями поставщика.
CD19-CD28 усиливает опосредованную CD20-TCB активацию Т-клеток на различных линиях В-клеток
Чтобы оценить способность антител к CD19-CD28 усиливать опосредованную CD20-TCB эффекторную функцию, оценивали активацию Т-клеток в комбинации с ТСВ. С этой целью репортерные клетки IL-2 Jurkat культивировали совместно с четырьмя разными экспрессирующими CD19 линиями клеток (Nalm6, WSU DLCL2, Z138, RCK8) в течение 6 ч в присутствии возрастающих концентраций CD19-CD28 v15 (Р1АЕ9040) и фиксированной ограничивающей концентрации CD20-TCB (P1AD4071). CD20 ТСВ представляет собой анти-CD20/анти-CD3 биспецифическое антитело в формате 2+1, описанное в примере 1 WO 2016/020309 А1. Как изображено на Фиг. 45А-45Г, CD19-CD28 повышает опосредованную CD20-TCB эффекторную функцию в присутствии всех четырех линий В-клеток зависимым от концентрации образом.
Связывающие CD28 варианты со сниженной аффинностью являются функциональными in vitro в биспецифическом формате CD19-CD28
Оригинальный клон CD28(SA) имеет аффинность KD=1 нМ. Высокоаффинные клоны антител подвержены риску периферических эффектов поглощения, в особенности если мишень на высоком уровне экспрессируется в периферической крови, как в случае CD28. Чтобы (i) снизить периферические эффекты поглощения и (ii) снизить риск периферической активации Т-клеток за счет связывания нацеленных, агонистов CD28 с Т-клетками за пределами опухоли, мы создали ряд из 31 клона CD28 со сниженной аффинностью путем внесения точечных мутаций в CDR (смотрите пример 1.1). Клоны-кандидатов выбирали так, как описано ранее. Создали 5 молекул с разной аффинностью к CD28 в нацеленном на CD19 биспецифическом формате: CD19-CD28 v8 (P1AF0175), CD19-CD28 v11 (P1AF0377), CD19-CD28 v15 (Р1АЕ9040), CD19-CD28 v27 (P1AF0378) и CD19-CD28 v29 (P1AE8002). На Фиг. 44А и 44Б показано, что все созданные молекулы CD19-CD28 связываются с CD19 человека, а также CD28, а интенсивность их связывания коррелирует с аффинностью связывающих молекул.
Чтобы оценить, были ли варианты клона CD28 со сниженной аффинностью функциональными и способными поддерживать ТСВ-опосредованные эффекторные функции, мы оценивали активацию Т-клеток в комбинации ТСВ. С этой целью репортерные IL-2 клетки Jurkat культивировали вместе с экспрессирующими CD19 клетками Nalm6 в течение 6 ч в присутствии возрастающих концентраций CD19-CD28 и фиксированной ограничивающей концентрацией CD20-TCB. Как изображено на Фиг. 46, все варианты связывающих CD28 молекул были функциональными и способными повышать ТСВ-опосредованную активацию Т-клеток зависимым от концентрации образом. Пан-Т-клетки использовали в качестве эффекторных клеток и выделяли из МКПК с помощью MACS, используя набор для выделения пан-Т-клеток (Miltenyi Biotec) в соответствии с инструкциями производителя.
CD19-CD28 не активирует Т-клетки МКПК в отвутствие сигналов ТКР
Чтобы подтвердить, что конструкции CD19-CD28 неактивны без сигнала ТКР, такого, который обеспечивает CD20-TCB, мы оценивали статус активации полученных из МКПК Т-клеток после совместного культивирования с экспрессирующими CD20 клетками-мишенями (Nalm6) и CD19-CD28 в отсутствие или присутствии CD20-TCB. Как изображено на Фиг. 47, CD19-CD28 повышает опосредованную CD20-TCB экспрессию CD69 в Т-клетках МКПК дозозависимым образом, тогда как в отсутствие ТСВ CD19-CD28 не приводит к активации Т-клеток даже при высоких концентрациях (100 нМ). Это открытие подтверждается нашим наблюдением, что CD19-CD28 не приводит к высвобождению цитокинов в полученных из МКПК Т-клетках после совместной инкубации с CD19-CD28 v8 или CD19-CD28 v15 в присутствии или отсутствие CD20-TCB (Фиг. 48А-48Г). В заключение, эти данные подтверждают, что CD19-CD28 не является суперагонистическим антителом, и для него необходим сигнал ТКР, т.е. обеспечиваемый ТСВ, для усиления активации Т-клеток.
CD79b-CD28 усиливает опосредованную CD20-TCB активацию Т-клеток В дополнение к нацеленным на CD19 агонистическим антителам к CD28 мы также создали нацеленные на CD79b антитела к CD28, чтобы оценить их способность усиливать опосредованную CD20-TCB активацию Т-клеток. Для этого использовали CD79b-положительную линию В-клеток Z138. Как показано на Фиг. 49А, CD79b-CD28 связывается с Z138. Кроме того, CD79b-CD28 было способно усиливать опосредованную CD20-TCB выработку IL-2 клетками IL-2 Jurkat при инкубации с Z138 в присутствии 0,05 нМ CD20-TCB (Фиг. 49Б).
Пример 22
In vivo функциональные характеристики биспецифических антигенсвязывающих молекул, нацеленных на CD28 и CD19
Исследование эффективности биспецифических антигенсвязывающих молекул к CD19-CD28 с разными вариантами CD28 в ксенотрансплантате NALM6 у гуманизированных мышей
Целью описанного в данном документе исследования эффективности была оценка того, какой вариант CD28 в биспецифической антигенсвязывающей молекуле CD19-CD28 приведет к более сильному ингибированию роста опухоли в монотерапии в модели CD19-положительной лимфомы человека у полностью гуманизированных мышей NSG.
Человеческие клетки NALM6 (лейкоз из предшественников В-клеток) были изначально получены от АТСС и после размножения помещены во внутренний банк клеток Glycart. Клетки культивировали в RPMI, содержащей 10% ФТС и 1x Glutamax. Клетки культивировали при 37°С в насыщенной влагой атмосфере при 5% СО2. 50 микролитров клеточной суспензии (1×106 клеток NALM6), смешанной с 50 микролитрами матригеля, подкожно инъецировали в бок анестезированных мышей с помощью 22G-30G иглы.
Самок мышей NSG возрастом 4 5 недель на момент начала эксперимента (Jackson Laboratory) содержали в специальных беспатогенных условиях с суточными циклами 12 ч день/12 ч ночь в соответствии с принятыми руководствами (GV-Solas; Felasa; TierschG). Протокол экспериментального исследования был рассмотрен и одобрен местными органами управления (ZH225-17). После поступления животных держали в течение одной недели для привыкания к новой среде и для наблюдения. Постоянное наблюдение за состоянием здоровья осуществляли на регулярной основе.
Самкам мышей NSG вводили в/б 15 мг/кг бусульфана, за чем через день следовала в/в инъекция 1×105 человеческих гемопоэтических клеток, выделенных из пуповинной крови. На 14 16 неделе после инъекции стволовых клеток у мышей сублингвально брали кровь и анализировали ее методом проточной цитометрии в отношении успешной гуманизации. Мышей с эффективным прививанием рандомизировали в соответствии с частотой человеческих Т-клеток в разные группы обработки. В это время мышам вводили п/к опухолевые клетки, как описано (Фиг. 50) и обрабатывали соединениями или гистидиновым буфером (носитель), когда размер опухолей достигал прибл. 150 мм3 (сутки 18). Всем мышам вводили в/в 200 мкл соответствующего раствора. Для получения надлежащего количества соединений на 200 мкл исходные растворы (таблица 44) разводили при необходимости гистидиновым буфером.

Рост опухоли измеряли дважды в неделю, используя штангенциркуль, а объем опухолей рассчитывали следующим образом:
Tv: (W2/2)×L (W: ширина, L: длина)
Исследование заканчивали на 53 сутки. На Фиг. 51А показана кинетика роста опухолей (среднее + СПС), а на Фиг. 51Б-51Г показана индивидуальная кинетика роста опухолей на группу и на мышь. Как описано в данном документе, вариант 8 CD19-CD28, как единственный агент, индуцировал более сильное ингибирование роста опухоли по сравнению с вариантом 15 CD19-CD28. Обработанное носителем животное пришлось умертвить раньше из-за образования метастазов после п/к введения опухолевых клеток. Монотерапевтический эффект у гуманизированных мышей можно объяснить усилением аллореактивных Т-клеток человека, которые можно рассматривать как суррогат для распознавания неоантигенов в мышиной системе.
Список литературы:
Acuto, О., and Michel, F. (2003). CD28-mediated co-stimulation: a quantitative support for TCR signalling. Nat Rev Immunol 3, 939-951.
Atamaniuk, J., Gleiss A., Porpazcy E., Kainz В., Grunt T.W., Raderer M., Hilgarth В., Drach J., Ludwig H., Gisslinger H., Jaeger U., Gaiger A. (2012). Overexpression of G protein-coupled receptor 5D in the bone marrow is associated with poor prognosis in patients with multiple myeloma. Eur J Clin Invest. 42(9), 953-60.
Bahlis N.J., King A.M., Kolonias D. Carlson L.M., Liu H.Y., Hussein M.A., Terebelo H.R., Byrne G.E. Jr, Levine B.L., Boise L.H., Lee K.P. (2007). CD28-mediated regulation of multiple myeloma cell proliferation and survival. Blood 109 (11), 5002-5010.
Boomer, J.S., and Green, J.M. (2010). An enigmatic tail of CD28 signaling. Cold Spring Harb Perspect Biol 2, a002436.
Brauner-Osborne H., Jensen A.A., Sheppard P.O., Brodin В., Krogsgaard-Larsen P., O'Hara P. (2001). Cloning and characterization of a human orphan family С G-protein coupled receptor GPRC5D. Biochim. Biophys. Acta 1518(3), 237-48.
Carreno, B.M., and Collins, M. (2002). The B7 family of ligands and its receptors: new pathways for costimulation and inhibition of immune responses. Annu Rev Immunol 20, 29-53.
Chen, L., and Flies, D.B. (2013). Molecular mechanisms of T cell co-stimulation and co-inhibition. Nat Rev Immunol 13, 227-242.
Cho S.F., Anderson K.C., Tai Y.T. (2018). Targeting В Cell Maturation Antigen (BCMA) in Multiple Myeloma: Potential Uses of BCMA-Based Immunotherapy. Front Immunol. 9, 1821.
Cohen Y., Gutwein O., Garach-Jehoshua O., Bar-Haim A., Kornberg A. (2013). GPRC5D is a promising marker for monitoring the tumor load and to target multiple myeloma cells. Hematology 18(6), 348-51.
Engelhardt, J.J., Sullivan, T.J., and Allison, J.P. (2006). CTLA-4 overexpression inhibits T cell responses through a CD28-B7-dependent mechanism. J Immunol 177, 1052-1061.
Esensten, J.H., Helou, Y.A., Chopra, G., Weiss, A., and Bluestone, J.A. (2016). CD28 Costimulation: From Mechanism to Therapy. Immunity 44, 973-988.
Fraser, J.D., Irving, B.A., Crabtree, G.R., and Weiss, A. (1991). Regulation of interleukin-2 gene enhancer activity by the T cell accessory molecule CD28. Science 251, 313-316.
Gao Y., Wang X., Yan H., Zeng J., Ma S., Niu Y., Zhou G., Jiang Y., Chen Y. (2016). Comparative Transcriptome Analysis of Fetal Skin Reveals Key Genes Related to Hair Follicle Morphogenesis in Cashmere Goats. PLoS One 11(3), e0151118.
Hui, E., Cheung, J., Zhu, J., Su, X., Taylor, M.J., Wallweber, H.A., Sasmal, D.K., Huang, J., Kim, J.M., Mellman, I., and Vale, R.D. (2017). T cell costimulatory receptor CD28 is a primary target for PD-1-mediated inhibition. Science 355, 1428-1433.
Hunig, T. (2012). The storm has cleared: lessons from the CD28 superagonist TGN1412 trial. Nat Rev Immunol 12, 317-318.
Inoue, S., Nambu T. and Shimomura T. (2004). The RAIG family member, GPRC5D, is associated with hard-keratinized structures. J Invest Dermatol. 122(3), 565-73.
June, C.H., Ledbetter, J.A., Gillespie, M.M., Lindsten, Т., and Thompson, C.B. (1987). T-cell proliferation involving the CD28 pathway is associated with cyclosporine-resistant interleukin 2 gene expression. Mol Cell Biol 7, 4472-4481.
Kamphorst, A.O., Wieland, A., Nasti, Т., Yang, S., Zhang, R., Barber, D.L., Konieczny, B.T., Daugherty, C.Z., Koenig, L., Yu, K., et al. (2017). Rescue of exhausted CD8 T cells by PD-1-targeted therapies is CD28-dependent. Science 355, 1423-1427.
Lavin, Y., Kobayashi, S., Leader, A., Amir, E.D., Elefant, N., Bigenwald, C., Remark, R., Sweeney, R., Becker, C.D., Levine, J.H., et al. (2017). Innate Immune Landscape in Early Lung Adenocarcinoma by Paired Single-Cell Analyses. Cell 169, 750-765 e717.
Linsley, P.S., Clark, E.A., and Ledbetter, J.A. (1990). T-cell antigen CD28 mediates adhesion with В cells by interacting with activation antigen B7/BB-1. Proc Natl Acad Sci USA 87, 5031-5035.
Moreau, P. and S.V. Rajkumar (2016). Multiple myeloma-translation of trial results into reality. Lancet, 388(10040): p. 111-113.
Murray M.E., Gavile С.М., Nair J.R., Koorella C., Carlson L.M., Buac D., Utley A., Chesi M., Bergsagel P.L., Boise L.H., Lee K.P. (2014). CD28-mediated pro-survival signaling induces chemotherapeutic resistance in multiple myeloma. Blood 123 (24), 3770-3779.
 Berr, S., Avota, E., Na, S.Y., Battaglia, M., ten Berge, I., Einsele, FL, and Hunig, T. (2011). Preculture of PBMCs at high cell density increases sensitivity of T-cell responses, revealing cytokine release by CD28 superagonist TGN1412. Blood 118, 6772-6782.
Berr, S., Avota, E., Na, S.Y., Battaglia, M., ten Berge, I., Einsele, FL, and Hunig, T. (2011). Preculture of PBMCs at high cell density increases sensitivity of T-cell responses, revealing cytokine release by CD28 superagonist TGN1412. Blood 118, 6772-6782.
Tai, X., Van Laethem, F., Sharpe, A.H., and Singer, A. (2007). Induction of autoimmune disease in CTLA-4-/- mice depends on a specific CD28 motif that is required for in vivo costimulation. Proc Natl Acad Sci USA 104, 13756-13761.
Thompson, C.B., Lindsten, Т., Ledbetter, J.A., Kunkel, S.L., Young, H.A., Emerson, S.G., Leiden, J.M., and June, C.H. (1989). CD28 activation pathway regulates the production of multiple T-cell-derived lymphokines/cytokines. Proc Natl Acad Sci USA 86, 1333-1337.
Tirosh, I., Izar, В., Prakadan, S.M., Wadsworth, M.H., 2nd, Treacy, D., Trombetta, J.J., Rotem, A., Rodman, C., Lian, C., Murphy, G., et al. (2016). Dissecting the multicellular ecosystem of metastatic melanoma by single-cell RNA-seq. Science 352, 189-196.
Zheng, C., Zheng, L., Yoo, J.K., Guo, H., Zhang, Y., Guo, X., Kang, В., Hu, R., Huang, J.Y., Zhang, Q., et al. (2017). Landscape of Infiltrating T Cells in Liver Cancer Revealed by Single-Cell Sequencing. Cell 169, 1342-1356 el316.
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> Ф.ХОФФМАНН-ЛЯ РОШ
<120> НАЦЕЛЕННЫЕ НА ОПУХОЛЬ АГОНИСТИЧЕСКИЕ CD28-АНТИГЕНСВЯЗЫВАЮЩИЕ
МОЛЕКУЛЫ
<130> P35128-WO
<150> EP18215121.7
<151> 2018-12-21
<150> EP19187709.1
<151> 2019-07-23
<150> EP19196006.1
<151> 2019-09-06
<160> 611
<170> PatentIn, версия 3.5
<210> 1
<211> 220
<212> Белок
<213> Homo sapiens
<400> 1
Met Leu Arg Leu Leu Leu Ala Leu Asn Leu Phe Pro Ser Ile Gln Val
1 5 10 15
Thr Gly Asn Lys Ile Leu Val Lys Gln Ser Pro Met Leu Val Ala Tyr
20 25 30
Asp Asn Ala Val Asn Leu Ser Cys Lys Tyr Ser Tyr Asn Leu Phe Ser
35 40 45
Arg Glu Phe Arg Ala Ser Leu His Lys Gly Leu Asp Ser Ala Val Glu
50 55 60
Val Cys Val Val Tyr Gly Asn Tyr Ser Gln Gln Leu Gln Val Tyr Ser
65 70 75 80
Lys Thr Gly Phe Asn Cys Asp Gly Lys Leu Gly Asn Glu Ser Val Thr
85 90 95
Phe Tyr Leu Gln Asn Leu Tyr Val Asn Gln Thr Asp Ile Tyr Phe Cys
100 105 110
Lys Ile Glu Val Met Tyr Pro Pro Pro Tyr Leu Asp Asn Glu Lys Ser
115 120 125
Asn Gly Thr Ile Ile His Val Lys Gly Lys His Leu Cys Pro Ser Pro
130 135 140
Leu Phe Pro Gly Pro Ser Lys Pro Phe Trp Val Leu Val Val Val Gly
145 150 155 160
Gly Val Leu Ala Cys Tyr Ser Leu Leu Val Thr Val Ala Phe Ile Ile
165 170 175
Phe Trp Val Arg Ser Lys Arg Ser Arg Leu Leu His Ser Asp Tyr Met
180 185 190
Asn Met Thr Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln Pro
195 200 205
Tyr Ala Pro Pro Arg Asp Phe Ala Ala Tyr Arg Ser
210 215 220
<210> 2
<211> 760
<212> Белок
<213> Homo sapiens
<400> 2
Met Lys Thr Trp Val Lys Ile Val Phe Gly Val Ala Thr Ser Ala Val
1 5 10 15
Leu Ala Leu Leu Val Met Cys Ile Val Leu Arg Pro Ser Arg Val His
20 25 30
Asn Ser Glu Glu Asn Thr Met Arg Ala Leu Thr Leu Lys Asp Ile Leu
35 40 45
Asn Gly Thr Phe Ser Tyr Lys Thr Phe Phe Pro Asn Trp Ile Ser Gly
50 55 60
Gln Glu Tyr Leu His Gln Ser Ala Asp Asn Asn Ile Val Leu Tyr Asn
65 70 75 80
Ile Glu Thr Gly Gln Ser Tyr Thr Ile Leu Ser Asn Arg Thr Met Lys
85 90 95
Ser Val Asn Ala Ser Asn Tyr Gly Leu Ser Pro Asp Arg Gln Phe Val
100 105 110
Tyr Leu Glu Ser Asp Tyr Ser Lys Leu Trp Arg Tyr Ser Tyr Thr Ala
115 120 125
Thr Tyr Tyr Ile Tyr Asp Leu Ser Asn Gly Glu Phe Val Arg Gly Asn
130 135 140
Glu Leu Pro Arg Pro Ile Gln Tyr Leu Cys Trp Ser Pro Val Gly Ser
145 150 155 160
Lys Leu Ala Tyr Val Tyr Gln Asn Asn Ile Tyr Leu Lys Gln Arg Pro
165 170 175
Gly Asp Pro Pro Phe Gln Ile Thr Phe Asn Gly Arg Glu Asn Lys Ile
180 185 190
Phe Asn Gly Ile Pro Asp Trp Val Tyr Glu Glu Glu Met Leu Ala Thr
195 200 205
Lys Tyr Ala Leu Trp Trp Ser Pro Asn Gly Lys Phe Leu Ala Tyr Ala
210 215 220
Glu Phe Asn Asp Thr Asp Ile Pro Val Ile Ala Tyr Ser Tyr Tyr Gly
225 230 235 240
Asp Glu Gln Tyr Pro Arg Thr Ile Asn Ile Pro Tyr Pro Lys Ala Gly
245 250 255
Ala Lys Asn Pro Val Val Arg Ile Phe Ile Ile Asp Thr Thr Tyr Pro
260 265 270
Ala Tyr Val Gly Pro Gln Glu Val Pro Val Pro Ala Met Ile Ala Ser
275 280 285
Ser Asp Tyr Tyr Phe Ser Trp Leu Thr Trp Val Thr Asp Glu Arg Val
290 295 300
Cys Leu Gln Trp Leu Lys Arg Val Gln Asn Val Ser Val Leu Ser Ile
305 310 315 320
Cys Asp Phe Arg Glu Asp Trp Gln Thr Trp Asp Cys Pro Lys Thr Gln
325 330 335
Glu His Ile Glu Glu Ser Arg Thr Gly Trp Ala Gly Gly Phe Phe Val
340 345 350
Ser Thr Pro Val Phe Ser Tyr Asp Ala Ile Ser Tyr Tyr Lys Ile Phe
355 360 365
Ser Asp Lys Asp Gly Tyr Lys His Ile His Tyr Ile Lys Asp Thr Val
370 375 380
Glu Asn Ala Ile Gln Ile Thr Ser Gly Lys Trp Glu Ala Ile Asn Ile
385 390 395 400
Phe Arg Val Thr Gln Asp Ser Leu Phe Tyr Ser Ser Asn Glu Phe Glu
405 410 415
Glu Tyr Pro Gly Arg Arg Asn Ile Tyr Arg Ile Ser Ile Gly Ser Tyr
420 425 430
Pro Pro Ser Lys Lys Cys Val Thr Cys His Leu Arg Lys Glu Arg Cys
435 440 445
Gln Tyr Tyr Thr Ala Ser Phe Ser Asp Tyr Ala Lys Tyr Tyr Ala Leu
450 455 460
Val Cys Tyr Gly Pro Gly Ile Pro Ile Ser Thr Leu His Asp Gly Arg
465 470 475 480
Thr Asp Gln Glu Ile Lys Ile Leu Glu Glu Asn Lys Glu Leu Glu Asn
485 490 495
Ala Leu Lys Asn Ile Gln Leu Pro Lys Glu Glu Ile Lys Lys Leu Glu
500 505 510
Val Asp Glu Ile Thr Leu Trp Tyr Lys Met Ile Leu Pro Pro Gln Phe
515 520 525
Asp Arg Ser Lys Lys Tyr Pro Leu Leu Ile Gln Val Tyr Gly Gly Pro
530 535 540
Cys Ser Gln Ser Val Arg Ser Val Phe Ala Val Asn Trp Ile Ser Tyr
545 550 555 560
Leu Ala Ser Lys Glu Gly Met Val Ile Ala Leu Val Asp Gly Arg Gly
565 570 575
Thr Ala Phe Gln Gly Asp Lys Leu Leu Tyr Ala Val Tyr Arg Lys Leu
580 585 590
Gly Val Tyr Glu Val Glu Asp Gln Ile Thr Ala Val Arg Lys Phe Ile
595 600 605
Glu Met Gly Phe Ile Asp Glu Lys Arg Ile Ala Ile Trp Gly Trp Ser
610 615 620
Tyr Gly Gly Tyr Val Ser Ser Leu Ala Leu Ala Ser Gly Thr Gly Leu
625 630 635 640
Phe Lys Cys Gly Ile Ala Val Ala Pro Val Ser Ser Trp Glu Tyr Tyr
645 650 655
Ala Ser Val Tyr Thr Glu Arg Phe Met Gly Leu Pro Thr Lys Asp Asp
660 665 670
Asn Leu Glu His Tyr Lys Asn Ser Thr Val Met Ala Arg Ala Glu Tyr
675 680 685
Phe Arg Asn Val Asp Tyr Leu Leu Ile His Gly Thr Ala Asp Asp Asn
690 695 700
Val His Phe Gln Asn Ser Ala Gln Ile Ala Lys Ala Leu Val Asn Ala
705 710 715 720
Gln Val Asp Phe Gln Ala Met Trp Tyr Ser Asp Gln Asn His Gly Leu
725 730 735
Ser Gly Leu Ser Thr Asn His Leu Tyr Thr His Met Thr His Phe Leu
740 745 750
Lys Gln Cys Phe Ser Leu Ser Asp
755 760
<210> 3
<211> 702
<212> Белок
<213> Homo sapiens
<400> 3
Met Glu Ser Pro Ser Ala Pro Pro His Arg Trp Cys Ile Pro Trp Gln
1 5 10 15
Arg Leu Leu Leu Thr Ala Ser Leu Leu Thr Phe Trp Asn Pro Pro Thr
20 25 30
Thr Ala Lys Leu Thr Ile Glu Ser Thr Pro Phe Asn Val Ala Glu Gly
35 40 45
Lys Glu Val Leu Leu Leu Val His Asn Leu Pro Gln His Leu Phe Gly
50 55 60
Tyr Ser Trp Tyr Lys Gly Glu Arg Val Asp Gly Asn Arg Gln Ile Ile
65 70 75 80
Gly Tyr Val Ile Gly Thr Gln Gln Ala Thr Pro Gly Pro Ala Tyr Ser
85 90 95
Gly Arg Glu Ile Ile Tyr Pro Asn Ala Ser Leu Leu Ile Gln Asn Ile
100 105 110
Ile Gln Asn Asp Thr Gly Phe Tyr Thr Leu His Val Ile Lys Ser Asp
115 120 125
Leu Val Asn Glu Glu Ala Thr Gly Gln Phe Arg Val Tyr Pro Glu Leu
130 135 140
Pro Lys Pro Ser Ile Ser Ser Asn Asn Ser Lys Pro Val Glu Asp Lys
145 150 155 160
Asp Ala Val Ala Phe Thr Cys Glu Pro Glu Thr Gln Asp Ala Thr Tyr
165 170 175
Leu Trp Trp Val Asn Asn Gln Ser Leu Pro Val Ser Pro Arg Leu Gln
180 185 190
Leu Ser Asn Gly Asn Arg Thr Leu Thr Leu Phe Asn Val Thr Arg Asn
195 200 205
Asp Thr Ala Ser Tyr Lys Cys Glu Thr Gln Asn Pro Val Ser Ala Arg
210 215 220
Arg Ser Asp Ser Val Ile Leu Asn Val Leu Tyr Gly Pro Asp Ala Pro
225 230 235 240
Thr Ile Ser Pro Leu Asn Thr Ser Tyr Arg Ser Gly Glu Asn Leu Asn
245 250 255
Leu Ser Cys His Ala Ala Ser Asn Pro Pro Ala Gln Tyr Ser Trp Phe
260 265 270
Val Asn Gly Thr Phe Gln Gln Ser Thr Gln Glu Leu Phe Ile Pro Asn
275 280 285
Ile Thr Val Asn Asn Ser Gly Ser Tyr Thr Cys Gln Ala His Asn Ser
290 295 300
Asp Thr Gly Leu Asn Arg Thr Thr Val Thr Thr Ile Thr Val Tyr Ala
305 310 315 320
Glu Pro Pro Lys Pro Phe Ile Thr Ser Asn Asn Ser Asn Pro Val Glu
325 330 335
Asp Glu Asp Ala Val Ala Leu Thr Cys Glu Pro Glu Ile Gln Asn Thr
340 345 350
Thr Tyr Leu Trp Trp Val Asn Asn Gln Ser Leu Pro Val Ser Pro Arg
355 360 365
Leu Gln Leu Ser Asn Asp Asn Arg Thr Leu Thr Leu Leu Ser Val Thr
370 375 380
Arg Asn Asp Val Gly Pro Tyr Glu Cys Gly Ile Gln Asn Lys Leu Ser
385 390 395 400
Val Asp His Ser Asp Pro Val Ile Leu Asn Val Leu Tyr Gly Pro Asp
405 410 415
Asp Pro Thr Ile Ser Pro Ser Tyr Thr Tyr Tyr Arg Pro Gly Val Asn
420 425 430
Leu Ser Leu Ser Cys His Ala Ala Ser Asn Pro Pro Ala Gln Tyr Ser
435 440 445
Trp Leu Ile Asp Gly Asn Ile Gln Gln His Thr Gln Glu Leu Phe Ile
450 455 460
Ser Asn Ile Thr Glu Lys Asn Ser Gly Leu Tyr Thr Cys Gln Ala Asn
465 470 475 480
Asn Ser Ala Ser Gly His Ser Arg Thr Thr Val Lys Thr Ile Thr Val
485 490 495
Ser Ala Glu Leu Pro Lys Pro Ser Ile Ser Ser Asn Asn Ser Lys Pro
500 505 510
Val Glu Asp Lys Asp Ala Val Ala Phe Thr Cys Glu Pro Glu Ala Gln
515 520 525
Asn Thr Thr Tyr Leu Trp Trp Val Asn Gly Gln Ser Leu Pro Val Ser
530 535 540
Pro Arg Leu Gln Leu Ser Asn Gly Asn Arg Thr Leu Thr Leu Phe Asn
545 550 555 560
Val Thr Arg Asn Asp Ala Arg Ala Tyr Val Cys Gly Ile Gln Asn Ser
565 570 575
Val Ser Ala Asn Arg Ser Asp Pro Val Thr Leu Asp Val Leu Tyr Gly
580 585 590
Pro Asp Thr Pro Ile Ile Ser Pro Pro Asp Ser Ser Tyr Leu Ser Gly
595 600 605
Ala Asn Leu Asn Leu Ser Cys His Ser Ala Ser Asn Pro Ser Pro Gln
610 615 620
Tyr Ser Trp Arg Ile Asn Gly Ile Pro Gln Gln His Thr Gln Val Leu
625 630 635 640
Phe Ile Ala Lys Ile Thr Pro Asn Asn Asn Gly Thr Tyr Ala Cys Phe
645 650 655
Val Ser Asn Leu Ala Thr Gly Arg Asn Asn Ser Ile Val Lys Ser Ile
660 665 670
Thr Val Ser Ala Ser Gly Thr Ser Pro Gly Leu Ser Ala Gly Ala Thr
675 680 685
Val Gly Ile Met Ile Gly Val Leu Val Gly Val Ala Leu Ile
690 695 700
<210> 4
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> FAP(28H1) CDR-H1
<400> 4
Ser His Ala Met Ser
1 5
<210> 5
<211> 16
<212> Белок
<213> Искусственная последовательность
<220>
<223> FAP(28H1) CDR-H2
<400> 5
Ala Ile Trp Ala Ser Gly Glu Gln Tyr Tyr Ala Asp Ser Val Lys Gly
1 5 10 15
<210> 6
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> FAP(28H1) CDR-H3
<400> 6
Gly Trp Leu Gly Asn Phe Asp Tyr
1 5
<210> 7
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> FAP(28H1) CDR-L1
<400> 7
Arg Ala Ser Gln Ser Val Ser Arg Ser Tyr Leu Ala
1 5 10
<210> 8
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> FAP(28H1) CDR-L2
<400> 8
Gly Ala Ser Thr Arg Ala Thr
1 5
<210> 9
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> FAP(28H1) CDR-L3
<400> 9
Gln Gln Gly Gln Val Ile Pro Pro Thr
1 5
<210> 10
<211> 116
<212> Белок
<213> Искусственная последовательность
<220>
<223> FAP(28H1) VH
<400> 10
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser His
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Trp Ala Ser Gly Glu Gln Tyr Tyr Ala Asp Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Lys Gly Trp Leu Gly Asn Phe Asp Tyr Trp Gly Gln Gly Thr Leu Val
100 105 110
Thr Val Ser Ser
115
<210> 11
<211> 108
<212> Белок
<213> Искусственная последовательность
<220>
<223> FAP(28H1) VL
<400> 11
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Arg Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Ile Gly Ala Ser Thr Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Gly Gln Val Ile Pro
85 90 95
Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 12
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> FAP(4B9) CDR-H1
<400> 12
Ser Tyr Ala Met Ser
1 5
<210> 13
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> FAP(4B9) CDR-H2
<400> 13
Ala Ile Ile Gly Ser Gly Ala Ser Thr Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 14
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> FAP(4B9) CDR-H3
<400> 14
Gly Trp Phe Gly Gly Phe Asn Tyr
1 5
<210> 15
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> FAP(4B9) CDR-L1
<400> 15
Arg Ala Ser Gln Ser Val Ser Arg Ser Tyr Leu Ala
1 5 10
<210> 16
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> FAP(4B9) CDR-L2
<400> 16
Val Gly Ser Arg Arg Ala Thr
1 5
<210> 17
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> FAP(4B9) CDR-L3
<400> 17
Gln Gln Gly Ile Met Leu Pro Pro Thr
1 5
<210> 18
<211> 117
<212> Белок
<213> Искусственная последовательность
<220>
<223> FAP(4B9) VH
<400> 18
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ile Gly Ser Gly Ala Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Gly Trp Phe Gly Gly Phe Asn Tyr Trp Gly Gln Gly Thr Leu
100 105 110
Val Thr Val Ser Ser
115
<210> 19
<211> 108
<212> Белок
<213> Искусственная последовательность
<220>
<223> FAP(4B9) VL
<400> 19
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Thr Ser Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Asn Val Gly Ser Arg Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Gly Ile Met Leu Pro
85 90 95
Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 20
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA) CDR-H1
<400> 20
Ser Tyr Tyr Ile His
1 5
<210> 21
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA) CDR-H2
<400> 21
Cys Ile Tyr Pro Gly Asn Val Asn Thr Asn Tyr Asn Glu Lys Phe Lys
1 5 10 15
Asp
<210> 22
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA) CDR-H3
<400> 22
Ser His Tyr Gly Leu Asp Trp Asn Phe Asp Val
1 5 10
<210> 23
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA) CDR-L1
<400> 23
His Ala Ser Gln Asn Ile Tyr Val Trp Leu Asn
1 5 10
<210> 24
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA) CDR-L2
<400> 24
Lys Ala Ser Asn Leu His Thr
1 5
<210> 25
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA) CDR-L3
<400> 25
Gln Gln Gly Gln Thr Tyr Pro Tyr Thr
1 5
<210> 26
<211> 120
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA) VH
<400> 26
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Cys Ile Tyr Pro Gly Asn Val Asn Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Trp Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 27
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA) VL
<400> 27
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys His Ala Ser Gln Asn Ile Tyr Val Trp
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gly Gln Thr Tyr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 28
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(mAb 9.3) CDR-H1
<400> 28
Asp Tyr Gly Val His
1 5
<210> 29
<211> 16
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(mAb 9.3) CDR-H2
<400> 29
Val Ile Trp Ala Gly Gly Gly Thr Asn Tyr Asn Ser Ala Leu Met Ser
1 5 10 15
<210> 30
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(mAb 9.3) CDR-H3
<400> 30
Asp Lys Gly Tyr Ser Tyr Tyr Tyr Ser Met Asp Tyr
1 5 10
<210> 31
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(mAb 9.3) CDR-L1
<400> 31
Arg Ala Ser Glu Ser Val Glu Tyr Tyr Val Thr Ser Leu Met Gln
1 5 10 15
<210> 32
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(mAb 9.3) CDR-L2
<400> 32
Ala Ala Ser Asn Val Glu Ser
1 5
<210> 33
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(mAb 9.3) CDR-L3
<400> 33
Gln Gln Ser Arg Lys Val Pro Tyr Thr
1 5
<210> 34
<211> 120
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(mAb 9.3) VH
<400> 34
Glu Val Lys Leu Gln Gln Ser Gly Pro Gly Leu Val Thr Pro Ser Gln
1 5 10 15
Ser Leu Ser Ile Thr Cys Thr Val Ser Gly Phe Ser Leu Ser Asp Tyr
20 25 30
Gly Val His Trp Val Arg Gln Ser Pro Gly Gln Gly Leu Glu Trp Leu
35 40 45
Gly Val Ile Trp Ala Gly Gly Gly Thr Asn Tyr Asn Ser Ala Leu Met
50 55 60
Ser Arg Lys Ser Ile Ser Lys Asp Asn Ser Lys Ser Gln Val Phe Leu
65 70 75 80
Lys Met Asn Ser Leu Gln Ala Asp Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Asp Lys Gly Tyr Ser Tyr Tyr Tyr Ser Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Ser Val Thr Val Ser Ser
115 120
<210> 35
<211> 111
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(mAb 9.3) VL
<400> 35
Asp Ile Glu Leu Thr Gln Ser Pro Ala Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Gln Arg Ala Thr Ile Ser Cys Arg Ala Ser Glu Ser Val Glu Tyr Tyr
20 25 30
Val Thr Ser Leu Met Gln Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro
35 40 45
Lys Leu Leu Ile Phe Ala Ala Ser Asn Val Glu Ser Gly Val Pro Ala
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asn Phe Ser Leu Asn Ile His
65 70 75 80
Pro Val Asp Glu Asp Asp Val Ala Met Tyr Phe Cys Gln Gln Ser Arg
85 90 95
Lys Val Pro Tyr Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105 110
<210> 36
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28 CDR-H1 консенсусная
<400> 36
Ser Tyr Tyr Ile His
1 5
<210> 37
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28 CDR-H2 консенсусная
<220>
<221> ВАРИАНТ
<222> (5)..(5)
<223> Gly или Arg
<220>
<221> ВАРИАНТ
<222> (6)..(6)
<223> Asn или Asp
<220>
<221> ВАРИАНТ
<222> (7)..(7)
<223> Val или Gly
<220>
<221> ВАРИАНТ
<222> (8)..(8)
<223> Asn, Gln или Ala
<400> 37
Ser Ile Tyr Pro Xaa Xaa Xaa Xaa Thr Asn Tyr Asn Glu Lys Phe Lys
1 5 10 15
Asp
<210> 38
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28 CDR-H3 консенсусная
<220>
<221> ВАРИАНТ
<222> (5)..(5)
<223> Leu или Ala
<220>
<221> ВАРИАНТ
<222> (7)..(7)
<223> Trp, His, Tyr или Phe
<400> 38
Ser His Tyr Gly Xaa Asp Xaa Asn Phe Asp Val
1 5 10
<210> 39
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28 CDR-L1 консенсусная
<220>
<221> ВАРИАНТ
<222> (1)..(1)
<223> His или Arg
<220>
<221> ВАРИАНТ
<222> (5)..(5)
<223> Asn или Gly
<220>
<221> ВАРИАНТ
<222> (7)..(7)
<223> Tyr или Ser
<220>
<221> ВАРИАНТ
<222> (8)..(8)
<223> Val или Asn
<220>
<221> ВАРИАНТ
<222> (9)..(9)
<223> Trp, His, Phe или Tyr
<400> 39
Xaa Ala Ser Gln Xaa Ile Xaa Xaa Xaa Leu Asn
1 5 10
<210> 40
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28 CDR-L2 консенсусная
<220>
<221> ВАРИАНТ
<222> (1)..(1)
<223> Lys или Tyr
<220>
<221> ВАРИАНТ
<222> (2)..(2)
<223> Ala или Gly
<220>
<221> ВАРИАНТ
<222> (4)..(4)
<223> Asn или Ser
<220>
<221> ВАРИАНТ
<222> (6)..(6)
<223> His или Tyr
<220>
<221> ВАРИАНТ
<222> (7)..(7)
<223> Thr или Ser
<400> 40
Xaa Xaa Ser Xaa Leu Xaa Xaa
1 5
<210> 41
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28 CDR-L3 консенсусная
<220>
<221> ВАРИАНТ
<222> (3)..(3)
<223> Gly или Ala
<400> 41
Gln Gln Xaa Gln Thr Tyr Pro Tyr Thr
1 5
<210> 42
<211> 120
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28 VH вариант a
<400> 42
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Ser Ile Tyr Pro Gly Asn Val Asn Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Trp Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 43
<211> 120
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28 VH вариант b
<400> 43
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Ser Ile Tyr Pro Gly Asn Val Gln Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp His Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 44
<211> 120
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28 VH вариант c
<400> 44
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Ser Ile Tyr Pro Gly Asn Val Gln Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Ala Asp His Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 45
<211> 120
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28 VH вариант d
<400> 45
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Ser Ile Tyr Pro Arg Asp Gly Gln Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Tyr Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 46
<211> 120
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28 VH вариант e
<400> 46
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Ser Ile Tyr Pro Gly Asn Val Gln Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Trp Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 47
<211> 120
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28 VH вариант f
<400> 47
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Ser Ile Tyr Pro Gly Asn Val Gln Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Phe Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 48
<211> 120
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28 VH вариант g
<400> 48
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Ser Ile Tyr Pro Arg Asn Val Gln Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp His Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 49
<211> 120
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28 VH вариант h
<400> 49
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Ser Ile Tyr Pro Arg Asp Val Gln Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp His Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 50
<211> 120
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28 VH вариант i
<400> 50
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Ser Ile Tyr Pro Gly Asn Val Asn Thr Arg Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Ala Asp Thr Ser Lys Asn Thr Ala Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Trp Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 51
<211> 120
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28 VH вариант j
<400> 51
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Ser Ile Tyr Pro Gly Asn Val Ala Thr Arg Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Ala Asp Thr Ser Lys Asn Thr Ala Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Trp Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 52
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28 VL вариант k
<400> 52
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys His Ala Ser Gln Asn Ile Tyr Val His
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ala Gln Thr Tyr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 53
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28 VL вариант l
<400> 53
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys His Ala Ser Gln Asn Ile Tyr Val Phe
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gly Gln Thr Tyr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 54
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28 VL вариант m
<400> 54
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys His Ala Ser Gln Asn Ile Tyr Val Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gly Gln Thr Tyr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 55
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28 VL вариант n
<400> 55
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys His Ala Ser Gln Gly Ile Ser Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gly Gln Thr Tyr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 56
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28 VL вариант o
<400> 56
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys His Ala Ser Gln Asn Ile Tyr Val Trp
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Tyr Thr Ser Ser Leu His Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gly Gln Thr Tyr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 57
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28 VL вариант p
<400> 57
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys His Ala Ser Gln Gly Ile Ser Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Tyr Thr Ser Ser Leu His Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gly Gln Thr Tyr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 58
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28 VL вариант q
<400> 58
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys His Ala Ser Gln Gly Ile Ser Asn His
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gly Gln Thr Tyr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 59
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28 VL вариант r
<400> 59
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys His Ala Ser Gln Gly Ile Tyr Val Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gly Gln Thr Tyr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 60
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28 VL вариант s
<400> 60
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys His Ala Ser Gln Gly Ile Ser Val Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gly Gln Thr Tyr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 61
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28 VL вариант t
<400> 61
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Asn Ile Tyr Val Trp
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu Tyr Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gly Gln Thr Tyr Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 62
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA) легкая цепь
<400> 62
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys His Ala Ser Gln Asn Ile Tyr Val Trp
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gly Gln Thr Tyr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 63
<211> 447
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA) hu IgG4 тяжелая цепь
<400> 63
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Cys Ile Tyr Pro Gly Asn Val Asn Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Trp Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val Asp His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Ser Lys Tyr Gly Pro
210 215 220
Pro Cys Pro Ser Cys Pro Ala Pro Glu Phe Leu Gly Gly Pro Ser Val
225 230 235 240
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
245 250 255
Pro Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu
260 265 270
Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
275 280 285
Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser
290 295 300
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
305 310 315 320
Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile
325 330 335
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
340 345 350
Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
355 360 365
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
370 375 380
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
385 390 395 400
Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg
405 410 415
Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
420 425 430
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys
435 440 445
<210> 64
<211> 450
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA) hu IgG1 PGLALA тяжелая цепь
<400> 64
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Cys Ile Tyr Pro Gly Asn Val Asn Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Trp Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Lys
450
<210> 65
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA) hu IgG легкая цепь "RK"
<400> 65
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys His Ala Ser Gln Asn Ile Tyr Val Trp
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gly Gln Thr Tyr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Arg Lys Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 66
<211> 448
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA) hu IgG1 PGLALA Fc-выступ
<400> 66
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Cys Ile Tyr Pro Gly Asn Val Asn Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Trp Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Glu Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Glu Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
<210> 67
<211> 438
<212> Белок
<213> Искусственная последовательность
<220>
<223> FAP(4B9) VL-CH hu IgG1 PGLALA Fc-впадина
<400> 67
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Thr Ser Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Asn Val Gly Ser Arg Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Gly Ile Met Leu Pro
85 90 95
Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Ser Ser Ala Ser
100 105 110
Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr
115 120 125
Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro
130 135 140
Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val
145 150 155 160
His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser
165 170 175
Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile
180 185 190
Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val
195 200 205
Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala
210 215 220
Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro
225 230 235 240
Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val
245 250 255
Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val
260 265 270
Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln
275 280 285
Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln
290 295 300
Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala
305 310 315 320
Leu Gly Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro
325 330 335
Arg Glu Pro Gln Val Cys Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr
340 345 350
Lys Asn Gln Val Ser Leu Ser Cys Ala Val Lys Gly Phe Tyr Pro Ser
355 360 365
Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr
370 375 380
Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Val
385 390 395 400
Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe
405 410 415
Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys
420 425 430
Ser Leu Ser Leu Ser Pro
435
<210> 68
<211> 224
<212> Белок
<213> Искусственная последовательность
<220>
<223> FAP(4B9) VH-C-каппа
<400> 68
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ile Gly Ser Gly Ala Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Gly Trp Phe Gly Gly Phe Asn Tyr Trp Gly Gln Gly Thr Leu
100 105 110
Val Thr Val Ser Ser Ala Ser Val Ala Ala Pro Ser Val Phe Ile Phe
115 120 125
Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys
130 135 140
Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val
145 150 155 160
Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln
165 170 175
Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser
180 185 190
Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His
195 200 205
Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215 220
<210> 69
<211> 820
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA) VHCH-VHCH Fc-выступ FAP(4B9) VH PGLALA
<400> 69
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Cys Ile Tyr Pro Gly Asn Val Asn Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Trp Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln Leu Val Gln
225 230 235 240
Ser Gly Ala Glu Val Lys Lys Pro Gly Ala Ser Val Lys Val Ser Cys
245 250 255
Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr Tyr Ile His Trp Val Arg
260 265 270
Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile Gly Cys Ile Tyr Pro Gly
275 280 285
Asn Val Asn Thr Asn Tyr Asn Glu Lys Phe Lys Asp Arg Ala Thr Leu
290 295 300
Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr Met Glu Leu Ser Arg Leu
305 310 315 320
Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys Thr Arg Ser His Tyr Gly
325 330 335
Leu Asp Trp Asn Phe Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val
340 345 350
Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser
355 360 365
Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys
370 375 380
Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu
385 390 395 400
Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu
405 410 415
Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr
420 425 430
Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val
435 440 445
Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro
450 455 460
Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe
465 470 475 480
Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val
485 490 495
Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe
500 505 510
Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro
515 520 525
Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr
530 535 540
Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val
545 550 555 560
Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala
565 570 575
Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Cys Arg
580 585 590
Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Trp Cys Leu Val Lys Gly
595 600 605
Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro
610 615 620
Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser
625 630 635 640
Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln
645 650 655
Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His
660 665 670
Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Gly Gly Gly Gly Ser
675 680 685
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu
690 695 700
Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser
705 710 715 720
Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr Ala
725 730 735
Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ser
740 745 750
Ala Ile Ile Gly Ser Gly Ala Ser Thr Tyr Tyr Ala Asp Ser Val Lys
755 760 765
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu
770 775 780
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
785 790 795 800
Lys Gly Trp Phe Gly Gly Phe Asn Tyr Trp Gly Gln Gly Thr Leu Val
805 810 815
Thr Val Ser Ser
820
<210> 70
<211> 811
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA) VHCH-VHCH Fc-впадина FAP(4B9) VL PGLALA
<400> 70
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Cys Ile Tyr Pro Gly Asn Val Asn Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Trp Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln Leu Val Gln
225 230 235 240
Ser Gly Ala Glu Val Lys Lys Pro Gly Ala Ser Val Lys Val Ser Cys
245 250 255
Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr Tyr Ile His Trp Val Arg
260 265 270
Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile Gly Cys Ile Tyr Pro Gly
275 280 285
Asn Val Asn Thr Asn Tyr Asn Glu Lys Phe Lys Asp Arg Ala Thr Leu
290 295 300
Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr Met Glu Leu Ser Arg Leu
305 310 315 320
Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys Thr Arg Ser His Tyr Gly
325 330 335
Leu Asp Trp Asn Phe Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val
340 345 350
Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser
355 360 365
Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys
370 375 380
Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu
385 390 395 400
Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu
405 410 415
Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr
420 425 430
Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val
435 440 445
Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro
450 455 460
Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe
465 470 475 480
Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val
485 490 495
Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe
500 505 510
Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro
515 520 525
Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr
530 535 540
Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val
545 550 555 560
Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala
565 570 575
Lys Gly Gln Pro Arg Glu Pro Gln Val Cys Thr Leu Pro Pro Ser Arg
580 585 590
Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Ser Cys Ala Val Lys Gly
595 600 605
Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro
610 615 620
Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser
625 630 635 640
Phe Phe Leu Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln
645 650 655
Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His
660 665 670
Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Gly Gly Gly Gly Ser
675 680 685
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu
690 695 700
Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly Glu
705 710 715 720
Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Thr Ser Ser Tyr
725 730 735
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
740 745 750
Asn Val Gly Ser Arg Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser Gly
755 760 765
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu Pro
770 775 780
Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Gly Ile Met Leu Pro Pro
785 790 795 800
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
805 810
<210> 71
<211> 586
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA) VHCH- Fc-выступ FAP(4B9) VH
<400> 71
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Cys Ile Tyr Pro Gly Asn Val Asn Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Trp Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
450 455 460
Gly Gly Gly Gly Ser Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu
465 470 475 480
Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe
485 490 495
Thr Phe Ser Ser Tyr Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys
500 505 510
Gly Leu Glu Trp Val Ser Ala Ile Ile Gly Ser Gly Ala Ser Thr Tyr
515 520 525
Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser
530 535 540
Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr
545 550 555 560
Ala Val Tyr Tyr Cys Ala Lys Gly Trp Phe Gly Gly Phe Asn Tyr Trp
565 570 575
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
580 585
<210> 72
<211> 577
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA) VHCH- Fc-впадина FAP(4B9) VL
<400> 72
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Cys Ile Tyr Pro Gly Asn Val Asn Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Trp Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Cys
340 345 350
Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Ser Cys Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
450 455 460
Gly Gly Gly Gly Ser Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu
465 470 475 480
Ser Leu Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln
485 490 495
Ser Val Thr Ser Ser Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
500 505 510
Ala Pro Arg Leu Leu Ile Asn Val Gly Ser Arg Arg Ala Thr Gly Ile
515 520 525
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
530 535 540
Ile Ser Arg Leu Glu Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln
545 550 555 560
Gly Ile Met Leu Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile
565 570 575
Lys
<210> 73
<211> 693
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA) VHCH "EE"- Fc PGLALA FAP(4B9) VHCL
<400> 73
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Cys Ile Tyr Pro Gly Asn Val Asn Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Trp Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Glu Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Glu Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
450 455 460
Gly Gly Gly Gly Ser Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu
465 470 475 480
Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe
485 490 495
Thr Phe Ser Ser Tyr Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys
500 505 510
Gly Leu Glu Trp Val Ser Ala Ile Ile Gly Ser Gly Ala Ser Thr Tyr
515 520 525
Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser
530 535 540
Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr
545 550 555 560
Ala Val Tyr Tyr Cys Ala Lys Gly Trp Phe Gly Gly Phe Asn Tyr Trp
565 570 575
Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Val Ala Ala Pro
580 585 590
Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr
595 600 605
Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys
610 615 620
Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu
625 630 635 640
Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser
645 650 655
Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala
660 665 670
Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe
675 680 685
Asn Arg Gly Glu Cys
690
<210> 74
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> FAP(4B9) VLCH1
<400> 74
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Thr Ser Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Asn Val Gly Ser Arg Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Gly Ile Met Leu Pro
85 90 95
Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Ser Ser Ala Ser
100 105 110
Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr
115 120 125
Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro
130 135 140
Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val
145 150 155 160
His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser
165 170 175
Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile
180 185 190
Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val
195 200 205
Glu Pro Lys Ser Cys Asp
210
<210> 75
<211> 668
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA) VLCH1- FAP(4B9) VHCH1 "EE"- Fc-выступ PGLALA
<400> 75
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys His Ala Ser Gln Asn Ile Tyr Val Trp
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gly Gln Thr Tyr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Ser Ser Ala Ser Thr
100 105 110
Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser
115 120 125
Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu
130 135 140
Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His
145 150 155 160
Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser
165 170 175
Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys
180 185 190
Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu
195 200 205
Pro Lys Ser Cys Asp Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu
210 215 220
Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser
225 230 235 240
Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr Ala
245 250 255
Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ser
260 265 270
Ala Ile Ile Gly Ser Gly Ala Ser Thr Tyr Tyr Ala Asp Ser Val Lys
275 280 285
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu
290 295 300
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
305 310 315 320
Lys Gly Trp Phe Gly Gly Phe Asn Tyr Trp Gly Gln Gly Thr Leu Val
325 330 335
Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala
340 345 350
Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu
355 360 365
Val Glu Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly
370 375 380
Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser
385 390 395 400
Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu
405 410 415
Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr
420 425 430
Lys Val Asp Glu Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr
435 440 445
Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe
450 455 460
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
465 470 475 480
Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val
485 490 495
Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
500 505 510
Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val
515 520 525
Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
530 535 540
Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr Ile Ser
545 550 555 560
Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro
565 570 575
Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Trp Cys Leu Val
580 585 590
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
595 600 605
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp
610 615 620
Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
625 630 635 640
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
645 650 655
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
660 665
<210> 76
<211> 445
<212> Белок
<213> Искусственная последовательность
<220>
<223> FAP(4B9) VHCH1 "EE"- Fc-впадина PGLALA
<400> 76
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ile Gly Ser Gly Ala Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Gly Trp Phe Gly Gly Phe Asn Tyr Trp Gly Gln Gly Thr Leu
100 105 110
Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu
115 120 125
Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys
130 135 140
Leu Val Glu Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser
145 150 155 160
Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser
165 170 175
Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser
180 185 190
Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn
195 200 205
Thr Lys Val Asp Glu Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His
210 215 220
Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val
225 230 235 240
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
245 250 255
Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu
260 265 270
Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
275 280 285
Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser
290 295 300
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
305 310 315 320
Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr Ile
325 330 335
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Cys Thr Leu Pro
340 345 350
Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Ser Cys Ala
355 360 365
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
370 375 380
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
385 390 395 400
Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val Asp Lys Ser Arg
405 410 415
Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
420 425 430
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
<210> 77
<211> 227
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA) VHCL
<400> 77
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Cys Ile Tyr Pro Gly Asn Val Asn Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Trp Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Val Ala Ala Pro Ser Val
115 120 125
Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser
130 135 140
Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln
145 150 155 160
Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val
165 170 175
Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu
180 185 190
Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu
195 200 205
Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg
210 215 220
Gly Glu Cys
225
<210> 78
<211> 215
<212> Белок
<213> Искусственная последовательность
<220>
<223> FAP(4B9) VLCL "RK"
<400> 78
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Thr Ser Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Asn Val Gly Ser Arg Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Gly Ile Met Leu Pro
85 90 95
Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala
100 105 110
Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Arg Lys Leu Lys Ser
115 120 125
Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu
130 135 140
Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser
145 150 155 160
Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu
165 170 175
Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val
180 185 190
Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys
195 200 205
Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 79
<211> 225
<212> Белок
<213> Искусственная последовательность
<220>
<223> Fc-впадина PGLALA
<400> 79
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly
1 5 10 15
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
20 25 30
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
35 40 45
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
50 55 60
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
65 70 75 80
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
85 90 95
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile
100 105 110
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
115 120 125
Cys Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser
130 135 140
Leu Ser Cys Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
145 150 155 160
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
165 170 175
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val
180 185 190
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
195 200 205
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
210 215 220
Pro
225
<210> 80
<211> 363
<212> Белок
<213> Искусственная последовательность
<220>
<223> Fc-выступ -FAP(4B9) VH
<400> 80
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly
1 5 10 15
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
20 25 30
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
35 40 45
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
50 55 60
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
65 70 75 80
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
85 90 95
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile
100 105 110
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
115 120 125
Tyr Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser
130 135 140
Leu Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
145 150 155 160
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
165 170 175
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
180 185 190
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
195 200 205
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
210 215 220
Pro Gly Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
225 230 235 240
Ser Gly Gly Gly Gly Ser Glu Val Gln Leu Leu Glu Ser Gly Gly Gly
245 250 255
Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
260 265 270
Phe Thr Phe Ser Ser Tyr Ala Met Ser Trp Val Arg Gln Ala Pro Gly
275 280 285
Lys Gly Leu Glu Trp Val Ser Ala Ile Ile Gly Ser Gly Ala Ser Thr
290 295 300
Tyr Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn
305 310 315 320
Ser Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp
325 330 335
Thr Ala Val Tyr Tyr Cys Ala Lys Gly Trp Phe Gly Gly Phe Asn Tyr
340 345 350
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
355 360
<210> 81
<211> 697
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA) VHCH1 "EE"- Fc PGLALA CEA(Medi-565) VHCL
<400> 81
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Cys Ile Tyr Pro Gly Asn Val Asn Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Trp Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Glu Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Glu Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
450 455 460
Gly Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu
465 470 475 480
Val Gln Pro Gly Arg Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe
485 490 495
Thr Val Ser Ser Tyr Trp Met His Trp Val Arg Gln Ala Pro Gly Lys
500 505 510
Gly Leu Glu Trp Val Gly Phe Ile Arg Asn Lys Ala Asn Gly Gly Thr
515 520 525
Thr Glu Tyr Ala Ala Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp
530 535 540
Asp Ser Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu
545 550 555 560
Asp Thr Ala Val Tyr Tyr Cys Ala Arg Asp Arg Gly Leu Arg Phe Tyr
565 570 575
Phe Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser
580 585 590
Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu
595 600 605
Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro
610 615 620
Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly
625 630 635 640
Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr
645 650 655
Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His
660 665 670
Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val
675 680 685
Thr Lys Ser Phe Asn Arg Gly Glu Cys
690 695
<210> 82
<211> 221
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA-VLCH1
<400> 82
Gln Ala Val Leu Thr Gln Pro Ala Ser Leu Ser Ala Ser Pro Gly Ala
1 5 10 15
Ser Ala Ser Leu Thr Cys Thr Leu Arg Arg Gly Ile Asn Val Gly Ala
20 25 30
Tyr Ser Ile Tyr Trp Tyr Gln Gln Lys Pro Gly Ser Pro Pro Gln Tyr
35 40 45
Leu Leu Arg Tyr Lys Ser Asp Ser Asp Lys Gln Gln Gly Ser Gly Val
50 55 60
Ser Ser Arg Phe Ser Ala Ser Lys Asp Ala Ser Ala Asn Ala Gly Ile
65 70 75 80
Leu Leu Ile Ser Gly Leu Gln Ser Glu Asp Glu Ala Asp Tyr Tyr Cys
85 90 95
Met Ile Trp His Ser Gly Ala Ser Ala Val Phe Gly Gly Gly Thr Lys
100 105 110
Leu Thr Val Leu Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
210 215 220
<210> 83
<211> 590
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA) VHCH1- Fc-выступ CEA VH
<400> 83
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Cys Ile Tyr Pro Gly Asn Val Asn Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Trp Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
450 455 460
Gly Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu
465 470 475 480
Val Gln Pro Gly Arg Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe
485 490 495
Thr Val Ser Ser Tyr Trp Met His Trp Val Arg Gln Ala Pro Gly Lys
500 505 510
Gly Leu Glu Trp Val Gly Phe Ile Arg Asn Lys Ala Asn Gly Gly Thr
515 520 525
Thr Glu Tyr Ala Ala Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp
530 535 540
Asp Ser Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu
545 550 555 560
Asp Thr Ala Val Tyr Tyr Cys Ala Arg Asp Arg Gly Leu Arg Phe Tyr
565 570 575
Phe Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
580 585 590
<210> 84
<211> 585
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA) VHCH1- Fc-впадина CEA VL
<400> 84
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Cys Ile Tyr Pro Gly Asn Val Asn Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Trp Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Cys
340 345 350
Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Ser Cys Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
450 455 460
Gly Gly Gly Gly Ser Gln Ala Val Leu Thr Gln Pro Ala Ser Leu Ser
465 470 475 480
Ala Ser Pro Gly Ala Ser Ala Ser Leu Thr Cys Thr Leu Arg Arg Gly
485 490 495
Ile Asn Val Gly Ala Tyr Ser Ile Tyr Trp Tyr Gln Gln Lys Pro Gly
500 505 510
Ser Pro Pro Gln Tyr Leu Leu Arg Tyr Lys Ser Asp Ser Asp Lys Gln
515 520 525
Gln Gly Ser Gly Val Ser Ser Arg Phe Ser Ala Ser Lys Asp Ala Ser
530 535 540
Ala Asn Ala Gly Ile Leu Leu Ile Ser Gly Leu Gln Ser Glu Asp Glu
545 550 555 560
Ala Asp Tyr Tyr Cys Met Ile Trp His Ser Gly Ala Ser Ala Val Phe
565 570 575
Gly Gly Gly Thr Lys Leu Thr Val Leu
580 585
<210> 85
<211> 448
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA) VHCH1 "EE"- Fc-впадина PGLALA HYRF
<400> 85
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Cys Ile Tyr Pro Gly Asn Val Asn Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Trp Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Glu Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Glu Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Cys
340 345 350
Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Ser Cys Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn Arg Phe Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
<210> 86
<211> 225
<212> Белок
<213> Искусственная последовательность
<220>
<223> Fc-выступ PGLALA
<400> 86
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly
1 5 10 15
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
20 25 30
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
35 40 45
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
50 55 60
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
65 70 75 80
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
85 90 95
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile
100 105 110
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
115 120 125
Tyr Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser
130 135 140
Leu Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
145 150 155 160
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
165 170 175
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
180 185 190
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
195 200 205
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
210 215 220
Pro
225
<210> 87
<211> 446
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA VL-CH hu IgG1 PGLALA Fc-впадина
<400> 87
Gln Ala Val Leu Thr Gln Pro Ala Ser Leu Ser Ala Ser Pro Gly Ala
1 5 10 15
Ser Ala Ser Leu Thr Cys Thr Leu Arg Arg Gly Ile Asn Val Gly Ala
20 25 30
Tyr Ser Ile Tyr Trp Tyr Gln Gln Lys Pro Gly Ser Pro Pro Gln Tyr
35 40 45
Leu Leu Arg Tyr Lys Ser Asp Ser Asp Lys Gln Gln Gly Ser Gly Val
50 55 60
Ser Ser Arg Phe Ser Ala Ser Lys Asp Ala Ser Ala Asn Ala Gly Ile
65 70 75 80
Leu Leu Ile Ser Gly Leu Gln Ser Glu Asp Glu Ala Asp Tyr Tyr Cys
85 90 95
Met Ile Trp His Ser Gly Ala Ser Ala Val Phe Gly Gly Gly Thr Lys
100 105 110
Leu Thr Val Leu Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
210 215 220
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser
225 230 235 240
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
245 250 255
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
260 265 270
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
275 280 285
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val
290 295 300
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
305 310 315 320
Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr
325 330 335
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Cys Thr Leu
340 345 350
Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Ser Cys
355 360 365
Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
370 375 380
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
385 390 395 400
Ser Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val Asp Lys Ser
405 410 415
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
420 425 430
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
<210> 88
<211> 228
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA VH-CL
<400> 88
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Val Ser Ser Tyr
20 25 30
Trp Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Phe Ile Arg Asn Lys Ala Asn Gly Gly Thr Thr Glu Tyr Ala Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Ala Arg Asp Arg Gly Leu Arg Phe Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Val Ala Ala Pro Ser
115 120 125
Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala
130 135 140
Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val
145 150 155 160
Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser
165 170 175
Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr
180 185 190
Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys
195 200 205
Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn
210 215 220
Arg Gly Glu Cys
225
<210> 89
<211> 218
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(mAb 9.3) легкая цепь
<400> 89
Asp Ile Glu Leu Thr Gln Ser Pro Ala Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Gln Arg Ala Thr Ile Ser Cys Arg Ala Ser Glu Ser Val Glu Tyr Tyr
20 25 30
Val Thr Ser Leu Met Gln Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro
35 40 45
Lys Leu Leu Ile Phe Ala Ala Ser Asn Val Glu Ser Gly Val Pro Ala
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asn Phe Ser Leu Asn Ile His
65 70 75 80
Pro Val Asp Glu Asp Asp Val Ala Met Tyr Phe Cys Gln Gln Ser Arg
85 90 95
Lys Val Pro Tyr Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Arg
100 105 110
Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln
115 120 125
Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr
130 135 140
Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser
145 150 155 160
Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr
165 170 175
Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys
180 185 190
His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro
195 200 205
Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 90
<211> 448
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(mAb 9.3) hu IgG1 PGLALA тяжелая цепь
<400> 90
Glu Val Lys Leu Gln Gln Ser Gly Pro Gly Leu Val Thr Pro Ser Gln
1 5 10 15
Ser Leu Ser Ile Thr Cys Thr Val Ser Gly Phe Ser Leu Ser Asp Tyr
20 25 30
Gly Val His Trp Val Arg Gln Ser Pro Gly Gln Gly Leu Glu Trp Leu
35 40 45
Gly Val Ile Trp Ala Gly Gly Gly Thr Asn Tyr Asn Ser Ala Leu Met
50 55 60
Ser Arg Lys Ser Ile Ser Lys Asp Asn Ser Lys Ser Gln Val Phe Leu
65 70 75 80
Lys Met Asn Ser Leu Gln Ala Asp Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Asp Lys Gly Tyr Ser Tyr Tyr Tyr Ser Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Ser Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
<210> 91
<211> 218
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(mAb 9.3) hu IgG легкая цепь "RK"
<400> 91
Asp Ile Glu Leu Thr Gln Ser Pro Ala Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Gln Arg Ala Thr Ile Ser Cys Arg Ala Ser Glu Ser Val Glu Tyr Tyr
20 25 30
Val Thr Ser Leu Met Gln Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro
35 40 45
Lys Leu Leu Ile Phe Ala Ala Ser Asn Val Glu Ser Gly Val Pro Ala
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asn Phe Ser Leu Asn Ile His
65 70 75 80
Pro Val Asp Glu Asp Asp Val Ala Met Tyr Phe Cys Gln Gln Ser Arg
85 90 95
Lys Val Pro Tyr Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Arg
100 105 110
Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Arg Lys
115 120 125
Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr
130 135 140
Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser
145 150 155 160
Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr
165 170 175
Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys
180 185 190
His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro
195 200 205
Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 92
<211> 448
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(mAb 9.3) hu IgG1 PGLALA Fc-выступ "EE"
<400> 92
Glu Val Lys Leu Gln Gln Ser Gly Pro Gly Leu Val Thr Pro Ser Gln
1 5 10 15
Ser Leu Ser Ile Thr Cys Thr Val Ser Gly Phe Ser Leu Ser Asp Tyr
20 25 30
Gly Val His Trp Val Arg Gln Ser Pro Gly Gln Gly Leu Glu Trp Leu
35 40 45
Gly Val Ile Trp Ala Gly Gly Gly Thr Asn Tyr Asn Ser Ala Leu Met
50 55 60
Ser Arg Lys Ser Ile Ser Lys Asp Asn Ser Lys Ser Gln Val Phe Leu
65 70 75 80
Lys Met Asn Ser Leu Gln Ala Asp Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Asp Lys Gly Tyr Ser Tyr Tyr Tyr Ser Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Ser Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Glu Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Glu Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
<210> 93
<211> 820
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(mAb 9.3) VHCH-VHCH Fc-выступ FAP(4B9) VH PGLALA
<400> 93
Glu Val Lys Leu Gln Gln Ser Gly Pro Gly Leu Val Thr Pro Ser Gln
1 5 10 15
Ser Leu Ser Ile Thr Cys Thr Val Ser Gly Phe Ser Leu Ser Asp Tyr
20 25 30
Gly Val His Trp Val Arg Gln Ser Pro Gly Gln Gly Leu Glu Trp Leu
35 40 45
Gly Val Ile Trp Ala Gly Gly Gly Thr Asn Tyr Asn Ser Ala Leu Met
50 55 60
Ser Arg Lys Ser Ile Ser Lys Asp Asn Ser Lys Ser Gln Val Phe Leu
65 70 75 80
Lys Met Asn Ser Leu Gln Ala Asp Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Asp Lys Gly Tyr Ser Tyr Tyr Tyr Ser Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Ser Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Val Lys Leu Gln Gln
225 230 235 240
Ser Gly Pro Gly Leu Val Thr Pro Ser Gln Ser Leu Ser Ile Thr Cys
245 250 255
Thr Val Ser Gly Phe Ser Leu Ser Asp Tyr Gly Val His Trp Val Arg
260 265 270
Gln Ser Pro Gly Gln Gly Leu Glu Trp Leu Gly Val Ile Trp Ala Gly
275 280 285
Gly Gly Thr Asn Tyr Asn Ser Ala Leu Met Ser Arg Lys Ser Ile Ser
290 295 300
Lys Asp Asn Ser Lys Ser Gln Val Phe Leu Lys Met Asn Ser Leu Gln
305 310 315 320
Ala Asp Asp Thr Ala Val Tyr Tyr Cys Ala Arg Asp Lys Gly Tyr Ser
325 330 335
Tyr Tyr Tyr Ser Met Asp Tyr Trp Gly Gln Gly Thr Ser Val Thr Val
340 345 350
Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser
355 360 365
Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys
370 375 380
Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu
385 390 395 400
Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu
405 410 415
Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr
420 425 430
Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val
435 440 445
Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro
450 455 460
Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe
465 470 475 480
Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val
485 490 495
Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe
500 505 510
Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro
515 520 525
Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr
530 535 540
Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val
545 550 555 560
Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala
565 570 575
Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Cys Arg
580 585 590
Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Trp Cys Leu Val Lys Gly
595 600 605
Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro
610 615 620
Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser
625 630 635 640
Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln
645 650 655
Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His
660 665 670
Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Gly Gly Gly Gly Ser
675 680 685
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu
690 695 700
Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser
705 710 715 720
Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr Ala
725 730 735
Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ser
740 745 750
Ala Ile Ile Gly Ser Gly Ala Ser Thr Tyr Tyr Ala Asp Ser Val Lys
755 760 765
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu
770 775 780
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
785 790 795 800
Lys Gly Trp Phe Gly Gly Phe Asn Tyr Trp Gly Gln Gly Thr Leu Val
805 810 815
Thr Val Ser Ser
820
<210> 94
<211> 811
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(mAb 9.3) VHCH-VHCH Fc-впадина FAP(4B9) VL PGLALA
<400> 94
Glu Val Lys Leu Gln Gln Ser Gly Pro Gly Leu Val Thr Pro Ser Gln
1 5 10 15
Ser Leu Ser Ile Thr Cys Thr Val Ser Gly Phe Ser Leu Ser Asp Tyr
20 25 30
Gly Val His Trp Val Arg Gln Ser Pro Gly Gln Gly Leu Glu Trp Leu
35 40 45
Gly Val Ile Trp Ala Gly Gly Gly Thr Asn Tyr Asn Ser Ala Leu Met
50 55 60
Ser Arg Lys Ser Ile Ser Lys Asp Asn Ser Lys Ser Gln Val Phe Leu
65 70 75 80
Lys Met Asn Ser Leu Gln Ala Asp Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Asp Lys Gly Tyr Ser Tyr Tyr Tyr Ser Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Ser Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Val Lys Leu Gln Gln
225 230 235 240
Ser Gly Pro Gly Leu Val Thr Pro Ser Gln Ser Leu Ser Ile Thr Cys
245 250 255
Thr Val Ser Gly Phe Ser Leu Ser Asp Tyr Gly Val His Trp Val Arg
260 265 270
Gln Ser Pro Gly Gln Gly Leu Glu Trp Leu Gly Val Ile Trp Ala Gly
275 280 285
Gly Gly Thr Asn Tyr Asn Ser Ala Leu Met Ser Arg Lys Ser Ile Ser
290 295 300
Lys Asp Asn Ser Lys Ser Gln Val Phe Leu Lys Met Asn Ser Leu Gln
305 310 315 320
Ala Asp Asp Thr Ala Val Tyr Tyr Cys Ala Arg Asp Lys Gly Tyr Ser
325 330 335
Tyr Tyr Tyr Ser Met Asp Tyr Trp Gly Gln Gly Thr Ser Val Thr Val
340 345 350
Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser
355 360 365
Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys
370 375 380
Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu
385 390 395 400
Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu
405 410 415
Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr
420 425 430
Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val
435 440 445
Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro
450 455 460
Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe
465 470 475 480
Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val
485 490 495
Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe
500 505 510
Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro
515 520 525
Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr
530 535 540
Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val
545 550 555 560
Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala
565 570 575
Lys Gly Gln Pro Arg Glu Pro Gln Val Cys Thr Leu Pro Pro Ser Arg
580 585 590
Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Ser Cys Ala Val Lys Gly
595 600 605
Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro
610 615 620
Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser
625 630 635 640
Phe Phe Leu Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln
645 650 655
Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His
660 665 670
Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Gly Gly Gly Gly Ser
675 680 685
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu
690 695 700
Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly Glu
705 710 715 720
Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Thr Ser Ser Tyr
725 730 735
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
740 745 750
Asn Val Gly Ser Arg Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser Gly
755 760 765
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu Pro
770 775 780
Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Gly Ile Met Leu Pro Pro
785 790 795 800
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
805 810
<210> 95
<211> 586
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(mAb 9.3) VHCH- Fc-выступ FAP(4B9) VH
<400> 95
Glu Val Lys Leu Gln Gln Ser Gly Pro Gly Leu Val Thr Pro Ser Gln
1 5 10 15
Ser Leu Ser Ile Thr Cys Thr Val Ser Gly Phe Ser Leu Ser Asp Tyr
20 25 30
Gly Val His Trp Val Arg Gln Ser Pro Gly Gln Gly Leu Glu Trp Leu
35 40 45
Gly Val Ile Trp Ala Gly Gly Gly Thr Asn Tyr Asn Ser Ala Leu Met
50 55 60
Ser Arg Lys Ser Ile Ser Lys Asp Asn Ser Lys Ser Gln Val Phe Leu
65 70 75 80
Lys Met Asn Ser Leu Gln Ala Asp Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Asp Lys Gly Tyr Ser Tyr Tyr Tyr Ser Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Ser Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
450 455 460
Gly Gly Gly Gly Ser Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu
465 470 475 480
Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe
485 490 495
Thr Phe Ser Ser Tyr Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys
500 505 510
Gly Leu Glu Trp Val Ser Ala Ile Ile Gly Ser Gly Ala Ser Thr Tyr
515 520 525
Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser
530 535 540
Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr
545 550 555 560
Ala Val Tyr Tyr Cys Ala Lys Gly Trp Phe Gly Gly Phe Asn Tyr Trp
565 570 575
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
580 585
<210> 96
<211> 577
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(mAb 9.3) VHCH- Fc-впадина FAP(4B9) VL
<400> 96
Glu Val Lys Leu Gln Gln Ser Gly Pro Gly Leu Val Thr Pro Ser Gln
1 5 10 15
Ser Leu Ser Ile Thr Cys Thr Val Ser Gly Phe Ser Leu Ser Asp Tyr
20 25 30
Gly Val His Trp Val Arg Gln Ser Pro Gly Gln Gly Leu Glu Trp Leu
35 40 45
Gly Val Ile Trp Ala Gly Gly Gly Thr Asn Tyr Asn Ser Ala Leu Met
50 55 60
Ser Arg Lys Ser Ile Ser Lys Asp Asn Ser Lys Ser Gln Val Phe Leu
65 70 75 80
Lys Met Asn Ser Leu Gln Ala Asp Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Asp Lys Gly Tyr Ser Tyr Tyr Tyr Ser Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Ser Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Cys
340 345 350
Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Ser Cys Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
450 455 460
Gly Gly Gly Gly Ser Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu
465 470 475 480
Ser Leu Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln
485 490 495
Ser Val Thr Ser Ser Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
500 505 510
Ala Pro Arg Leu Leu Ile Asn Val Gly Ser Arg Arg Ala Thr Gly Ile
515 520 525
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
530 535 540
Ile Ser Arg Leu Glu Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln
545 550 555 560
Gly Ile Met Leu Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile
565 570 575
Lys
<210> 97
<211> 693
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(mAb 9.3) VHCH "EE"- Fc PGLALA FAP(4B9) VHCL
<400> 97
Glu Val Lys Leu Gln Gln Ser Gly Pro Gly Leu Val Thr Pro Ser Gln
1 5 10 15
Ser Leu Ser Ile Thr Cys Thr Val Ser Gly Phe Ser Leu Ser Asp Tyr
20 25 30
Gly Val His Trp Val Arg Gln Ser Pro Gly Gln Gly Leu Glu Trp Leu
35 40 45
Gly Val Ile Trp Ala Gly Gly Gly Thr Asn Tyr Asn Ser Ala Leu Met
50 55 60
Ser Arg Lys Ser Ile Ser Lys Asp Asn Ser Lys Ser Gln Val Phe Leu
65 70 75 80
Lys Met Asn Ser Leu Gln Ala Asp Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Asp Lys Gly Tyr Ser Tyr Tyr Tyr Ser Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Ser Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Glu Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Glu Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
450 455 460
Gly Gly Gly Gly Ser Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu
465 470 475 480
Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe
485 490 495
Thr Phe Ser Ser Tyr Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys
500 505 510
Gly Leu Glu Trp Val Ser Ala Ile Ile Gly Ser Gly Ala Ser Thr Tyr
515 520 525
Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser
530 535 540
Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr
545 550 555 560
Ala Val Tyr Tyr Cys Ala Lys Gly Trp Phe Gly Gly Phe Asn Tyr Trp
565 570 575
Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Val Ala Ala Pro
580 585 590
Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr
595 600 605
Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys
610 615 620
Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu
625 630 635 640
Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser
645 650 655
Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala
660 665 670
Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe
675 680 685
Asn Arg Gly Glu Cys
690
<210> 98
<211> 218
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(mAb 9.3) VLCL "RK"
<400> 98
Asp Ile Glu Leu Thr Gln Ser Pro Ala Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Gln Arg Ala Thr Ile Ser Cys Arg Ala Ser Glu Ser Val Glu Tyr Tyr
20 25 30
Val Thr Ser Leu Met Gln Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro
35 40 45
Lys Leu Leu Ile Phe Ala Ala Ser Asn Val Glu Ser Gly Val Pro Ala
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asn Phe Ser Leu Asn Ile His
65 70 75 80
Pro Val Asp Glu Asp Asp Val Ala Met Tyr Phe Cys Gln Gln Ser Arg
85 90 95
Lys Val Pro Tyr Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Arg
100 105 110
Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Arg Lys
115 120 125
Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr
130 135 140
Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser
145 150 155 160
Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr
165 170 175
Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys
180 185 190
His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro
195 200 205
Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 99
<211> 213
<212> Белок
<213> Искусственная последовательность
<220>
<223> FAP(4B9) VL-CH1
<400> 99
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Thr Ser Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Asn Val Gly Ser Arg Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Gly Ile Met Leu Pro
85 90 95
Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Ser Ser Ala Ser
100 105 110
Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr
115 120 125
Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro
130 135 140
Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val
145 150 155 160
His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser
165 170 175
Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile
180 185 190
Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val
195 200 205
Glu Pro Lys Ser Cys
210
<210> 100
<211> 672
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(mAb 9.3) VLCH1- FAP(4B9) VHCH1 "EE"- Fc-выступ PGLALA
<400> 100
Asp Ile Glu Leu Thr Gln Ser Pro Ala Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Gln Arg Ala Thr Ile Ser Cys Arg Ala Ser Glu Ser Val Glu Tyr Tyr
20 25 30
Val Thr Ser Leu Met Gln Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro
35 40 45
Lys Leu Leu Ile Phe Ala Ala Ser Asn Val Glu Ser Gly Val Pro Ala
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asn Phe Ser Leu Asn Ile His
65 70 75 80
Pro Val Asp Glu Asp Asp Val Ala Met Tyr Phe Cys Gln Gln Ser Arg
85 90 95
Lys Val Pro Tyr Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Ser
100 105 110
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser
115 120 125
Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
130 135 140
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
145 150 155 160
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
165 170 175
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln
180 185 190
Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp
195 200 205
Lys Lys Val Glu Pro Lys Ser Cys Asp Gly Gly Gly Gly Ser Gly Gly
210 215 220
Gly Gly Ser Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln
225 230 235 240
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
245 250 255
Ser Ser Tyr Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
260 265 270
Glu Trp Val Ser Ala Ile Ile Gly Ser Gly Ala Ser Thr Tyr Tyr Ala
275 280 285
Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn
290 295 300
Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
305 310 315 320
Tyr Tyr Cys Ala Lys Gly Trp Phe Gly Gly Phe Asn Tyr Trp Gly Gln
325 330 335
Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
340 345 350
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
355 360 365
Leu Gly Cys Leu Val Glu Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
370 375 380
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
385 390 395 400
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
405 410 415
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
420 425 430
Pro Ser Asn Thr Lys Val Asp Glu Lys Val Glu Pro Lys Ser Cys Asp
435 440 445
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
450 455 460
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
465 470 475 480
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
485 490 495
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
500 505 510
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
515 520 525
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
530 535 540
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
545 550 555 560
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
565 570 575
Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
580 585 590
Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
595 600 605
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
610 615 620
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
625 630 635 640
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
645 650 655
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
660 665 670
<210> 101
<211> 227
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(mAb 9.3) VHCL
<400> 101
Glu Val Lys Leu Gln Gln Ser Gly Pro Gly Leu Val Thr Pro Ser Gln
1 5 10 15
Ser Leu Ser Ile Thr Cys Thr Val Ser Gly Phe Ser Leu Ser Asp Tyr
20 25 30
Gly Val His Trp Val Arg Gln Ser Pro Gly Gln Gly Leu Glu Trp Leu
35 40 45
Gly Val Ile Trp Ala Gly Gly Gly Thr Asn Tyr Asn Ser Ala Leu Met
50 55 60
Ser Arg Lys Ser Ile Ser Lys Asp Asn Ser Lys Ser Gln Val Phe Leu
65 70 75 80
Lys Met Asn Ser Leu Gln Ala Asp Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Asp Lys Gly Tyr Ser Tyr Tyr Tyr Ser Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Ser Val Thr Val Ser Ser Ala Ser Val Ala Ala Pro Ser Val
115 120 125
Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser
130 135 140
Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln
145 150 155 160
Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val
165 170 175
Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu
180 185 190
Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu
195 200 205
Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg
210 215 220
Gly Glu Cys
225
<210> 102
<211> 697
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(mAb 9.3) VHCH1 "EE"- Fc PGLALA CEA VHCL
<400> 102
Glu Val Lys Leu Gln Gln Ser Gly Pro Gly Leu Val Thr Pro Ser Gln
1 5 10 15
Ser Leu Ser Ile Thr Cys Thr Val Ser Gly Phe Ser Leu Ser Asp Tyr
20 25 30
Gly Val His Trp Val Arg Gln Ser Pro Gly Gln Gly Leu Glu Trp Leu
35 40 45
Gly Val Ile Trp Ala Gly Gly Gly Thr Asn Tyr Asn Ser Ala Leu Met
50 55 60
Ser Arg Lys Ser Ile Ser Lys Asp Asn Ser Lys Ser Gln Val Phe Leu
65 70 75 80
Lys Met Asn Ser Leu Gln Ala Asp Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Asp Lys Gly Tyr Ser Tyr Tyr Tyr Ser Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Ser Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Glu Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Glu Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
450 455 460
Gly Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu
465 470 475 480
Val Gln Pro Gly Arg Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe
485 490 495
Thr Val Ser Ser Tyr Trp Met His Trp Val Arg Gln Ala Pro Gly Lys
500 505 510
Gly Leu Glu Trp Val Gly Phe Ile Arg Asn Lys Ala Asn Gly Gly Thr
515 520 525
Thr Glu Tyr Ala Ala Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp
530 535 540
Asp Ser Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu
545 550 555 560
Asp Thr Ala Val Tyr Tyr Cys Ala Arg Asp Arg Gly Leu Arg Phe Tyr
565 570 575
Phe Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser
580 585 590
Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu
595 600 605
Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro
610 615 620
Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly
625 630 635 640
Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr
645 650 655
Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His
660 665 670
Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val
675 680 685
Thr Lys Ser Phe Asn Arg Gly Glu Cys
690 695
<210> 103
<211> 590
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(mAb 9.3) VHCH1- Fc-выступ CEA VH
<400> 103
Glu Val Lys Leu Gln Gln Ser Gly Pro Gly Leu Val Thr Pro Ser Gln
1 5 10 15
Ser Leu Ser Ile Thr Cys Thr Val Ser Gly Phe Ser Leu Ser Asp Tyr
20 25 30
Gly Val His Trp Val Arg Gln Ser Pro Gly Gln Gly Leu Glu Trp Leu
35 40 45
Gly Val Ile Trp Ala Gly Gly Gly Thr Asn Tyr Asn Ser Ala Leu Met
50 55 60
Ser Arg Lys Ser Ile Ser Lys Asp Asn Ser Lys Ser Gln Val Phe Leu
65 70 75 80
Lys Met Asn Ser Leu Gln Ala Asp Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Asp Lys Gly Tyr Ser Tyr Tyr Tyr Ser Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Ser Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
450 455 460
Gly Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu
465 470 475 480
Val Gln Pro Gly Arg Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe
485 490 495
Thr Val Ser Ser Tyr Trp Met His Trp Val Arg Gln Ala Pro Gly Lys
500 505 510
Gly Leu Glu Trp Val Gly Phe Ile Arg Asn Lys Ala Asn Gly Gly Thr
515 520 525
Thr Glu Tyr Ala Ala Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp
530 535 540
Asp Ser Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu
545 550 555 560
Asp Thr Ala Val Tyr Tyr Cys Ala Arg Asp Arg Gly Leu Arg Phe Tyr
565 570 575
Phe Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
580 585 590
<210> 104
<211> 585
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(mAb 9.3) VHCH1- Fc-впадина CEA VL
<400> 104
Glu Val Lys Leu Gln Gln Ser Gly Pro Gly Leu Val Thr Pro Ser Gln
1 5 10 15
Ser Leu Ser Ile Thr Cys Thr Val Ser Gly Phe Ser Leu Ser Asp Tyr
20 25 30
Gly Val His Trp Val Arg Gln Ser Pro Gly Gln Gly Leu Glu Trp Leu
35 40 45
Gly Val Ile Trp Ala Gly Gly Gly Thr Asn Tyr Asn Ser Ala Leu Met
50 55 60
Ser Arg Lys Ser Ile Ser Lys Asp Asn Ser Lys Ser Gln Val Phe Leu
65 70 75 80
Lys Met Asn Ser Leu Gln Ala Asp Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Asp Lys Gly Tyr Ser Tyr Tyr Tyr Ser Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Ser Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Cys
340 345 350
Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Ser Cys Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
450 455 460
Gly Gly Gly Gly Ser Gln Ala Val Leu Thr Gln Pro Ala Ser Leu Ser
465 470 475 480
Ala Ser Pro Gly Ala Ser Ala Ser Leu Thr Cys Thr Leu Arg Arg Gly
485 490 495
Ile Asn Val Gly Ala Tyr Ser Ile Tyr Trp Tyr Gln Gln Lys Pro Gly
500 505 510
Ser Pro Pro Gln Tyr Leu Leu Arg Tyr Lys Ser Asp Ser Asp Lys Gln
515 520 525
Gln Gly Ser Gly Val Ser Ser Arg Phe Ser Ala Ser Lys Asp Ala Ser
530 535 540
Ala Asn Ala Gly Ile Leu Leu Ile Ser Gly Leu Gln Ser Glu Asp Glu
545 550 555 560
Ala Asp Tyr Tyr Cys Met Ile Trp His Ser Gly Ala Ser Ala Val Phe
565 570 575
Gly Gly Gly Thr Lys Leu Thr Val Leu
580 585
<210> 105
<211> 448
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(mAb 9.3) VHCH1 "EE"- Fc-впадина PGLALA HYRF
<400> 105
Glu Val Lys Leu Gln Gln Ser Gly Pro Gly Leu Val Thr Pro Ser Gln
1 5 10 15
Ser Leu Ser Ile Thr Cys Thr Val Ser Gly Phe Ser Leu Ser Asp Tyr
20 25 30
Gly Val His Trp Val Arg Gln Ser Pro Gly Gln Gly Leu Glu Trp Leu
35 40 45
Gly Val Ile Trp Ala Gly Gly Gly Thr Asn Tyr Asn Ser Ala Leu Met
50 55 60
Ser Arg Lys Ser Ile Ser Lys Asp Asn Ser Lys Ser Gln Val Phe Leu
65 70 75 80
Lys Met Asn Ser Leu Gln Ala Asp Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Asp Lys Gly Tyr Ser Tyr Tyr Tyr Ser Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Ser Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Glu Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Glu Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Cys
340 345 350
Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Ser Cys Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
<210> 106
<211> 218
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(mAb 9.3) VLCL "RK"
<400> 106
Asp Ile Glu Leu Thr Gln Ser Pro Ala Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Gln Arg Ala Thr Ile Ser Cys Arg Ala Ser Glu Ser Val Glu Tyr Tyr
20 25 30
Val Thr Ser Leu Met Gln Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro
35 40 45
Lys Leu Leu Ile Phe Ala Ala Ser Asn Val Glu Ser Gly Val Pro Ala
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asn Phe Ser Leu Asn Ile His
65 70 75 80
Pro Val Asp Glu Asp Asp Val Ala Met Tyr Phe Cys Gln Gln Ser Arg
85 90 95
Lys Val Pro Tyr Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Arg
100 105 110
Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Arg Lys
115 120 125
Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr
130 135 140
Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser
145 150 155 160
Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr
165 170 175
Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys
180 185 190
His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro
195 200 205
Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 107
<211> 834
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA) VHCH1 "EE" Fc-впадина PGLALA FAP(4B9) VH -
CEA(Medi-565)
VHCL
<400> 107
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Cys Ile Tyr Pro Gly Asn Val Asn Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Trp Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Glu Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Glu Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
450 455 460
Gly Gly Gly Gly Ser Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu
465 470 475 480
Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe
485 490 495
Thr Phe Ser Ser Tyr Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys
500 505 510
Gly Leu Glu Trp Val Ser Ala Ile Ile Gly Ser Gly Ala Ser Thr Tyr
515 520 525
Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser
530 535 540
Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr
545 550 555 560
Ala Val Tyr Tyr Cys Ala Lys Gly Trp Phe Gly Gly Phe Asn Tyr Trp
565 570 575
Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly
580 585 590
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Val
595 600 605
Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg Ser Leu
610 615 620
Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Val Ser Ser Tyr Trp Met
625 630 635 640
His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Gly Phe
645 650 655
Ile Arg Asn Lys Ala Asn Gly Gly Thr Thr Glu Tyr Ala Ala Ser Val
660 665 670
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr Leu Tyr
675 680 685
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
690 695 700
Ala Arg Asp Arg Gly Leu Arg Phe Tyr Phe Asp Tyr Trp Gly Gln Gly
705 710 715 720
Thr Thr Val Thr Val Ser Ser Ala Ser Val Ala Ala Pro Ser Val Phe
725 730 735
Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val
740 745 750
Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp
755 760 765
Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr
770 775 780
Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr
785 790 795 800
Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val
805 810 815
Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly
820 825 830
Glu Cys
<210> 108
<211> 577
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA) VHCH1 "EE" Fc-выступ PGLALA FAP(4B9) VL
<400> 108
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Cys Ile Tyr Pro Gly Asn Val Asn Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Trp Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Glu Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Glu Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Cys
340 345 350
Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Ser Cys Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
450 455 460
Gly Gly Gly Gly Ser Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu
465 470 475 480
Ser Leu Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln
485 490 495
Ser Val Thr Ser Ser Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
500 505 510
Ala Pro Arg Leu Leu Ile Asn Val Gly Ser Arg Arg Ala Thr Gly Ile
515 520 525
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
530 535 540
Ile Ser Arg Leu Glu Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln
545 550 555 560
Gly Ile Met Leu Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile
565 570 575
Lys
<210> 109
<211> 221
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA VLCH1
<400> 109
Gln Ala Val Leu Thr Gln Pro Ala Ser Leu Ser Ala Ser Pro Gly Ala
1 5 10 15
Ser Ala Ser Leu Thr Cys Thr Leu Arg Arg Gly Ile Asn Val Gly Ala
20 25 30
Tyr Ser Ile Tyr Trp Tyr Gln Gln Lys Pro Gly Ser Pro Pro Gln Tyr
35 40 45
Leu Leu Arg Tyr Lys Ser Asp Ser Asp Lys Gln Gln Gly Ser Gly Val
50 55 60
Ser Ser Arg Phe Ser Ala Ser Lys Asp Ala Ser Ala Asn Ala Gly Ile
65 70 75 80
Leu Leu Ile Ser Gly Leu Gln Ser Glu Asp Glu Ala Asp Tyr Tyr Cys
85 90 95
Met Ile Trp His Ser Gly Ala Ser Ala Val Phe Gly Gly Gly Thr Lys
100 105 110
Leu Thr Val Leu Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
210 215 220
<210> 110
<211> 727
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA) VHCH1 Fc-впадина PGLALA FAP(4B9) VH - CEA VH
<400> 110
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Cys Ile Tyr Pro Gly Asn Val Asn Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Trp Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
450 455 460
Gly Gly Gly Gly Ser Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu
465 470 475 480
Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe
485 490 495
Thr Phe Ser Ser Tyr Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys
500 505 510
Gly Leu Glu Trp Val Ser Ala Ile Ile Gly Ser Gly Ala Ser Thr Tyr
515 520 525
Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser
530 535 540
Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr
545 550 555 560
Ala Val Tyr Tyr Cys Ala Lys Gly Trp Phe Gly Gly Phe Asn Tyr Trp
565 570 575
Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly
580 585 590
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Val
595 600 605
Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg Ser Leu
610 615 620
Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Val Ser Ser Tyr Trp Met
625 630 635 640
His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Gly Phe
645 650 655
Ile Arg Asn Lys Ala Asn Gly Gly Thr Thr Glu Tyr Ala Ala Ser Val
660 665 670
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr Leu Tyr
675 680 685
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
690 695 700
Ala Arg Asp Arg Gly Leu Arg Phe Tyr Phe Asp Tyr Trp Gly Gln Gly
705 710 715 720
Thr Thr Val Thr Val Ser Ser
725
<210> 111
<211> 713
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA) VHCH1 Fc-выступ PGLALA FAP(4B9) VL - CEA VL
<400> 111
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Cys Ile Tyr Pro Gly Asn Val Asn Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Trp Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Cys
340 345 350
Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Ser Cys Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
450 455 460
Gly Gly Gly Gly Ser Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu
465 470 475 480
Ser Leu Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln
485 490 495
Ser Val Thr Ser Ser Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
500 505 510
Ala Pro Arg Leu Leu Ile Asn Val Gly Ser Arg Arg Ala Thr Gly Ile
515 520 525
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
530 535 540
Ile Ser Arg Leu Glu Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln
545 550 555 560
Gly Ile Met Leu Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile
565 570 575
Lys Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
580 585 590
Gly Gly Gly Gly Ser Gln Ala Val Leu Thr Gln Pro Ala Ser Leu Ser
595 600 605
Ala Ser Pro Gly Ala Ser Ala Ser Leu Thr Cys Thr Leu Arg Arg Gly
610 615 620
Ile Asn Val Gly Ala Tyr Ser Ile Tyr Trp Tyr Gln Gln Lys Pro Gly
625 630 635 640
Ser Pro Pro Gln Tyr Leu Leu Arg Tyr Lys Ser Asp Ser Asp Lys Gln
645 650 655
Gln Gly Ser Gly Val Ser Ser Arg Phe Ser Ala Ser Lys Asp Ala Ser
660 665 670
Ala Asn Ala Gly Ile Leu Leu Ile Ser Gly Leu Gln Ser Glu Asp Glu
675 680 685
Ala Asp Tyr Tyr Cys Met Ile Trp His Ser Gly Ala Ser Ala Val Phe
690 695 700
Gly Gly Gly Thr Lys Leu Thr Val Leu
705 710
<210> 112
<211> 448
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH (родительская CD28) CH1- Fc-выступ PGLALA
<400> 112
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Cys Ile Tyr Pro Gly Asn Val Asn Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Trp Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Glu Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Glu Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
<210> 113
<211> 448
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH (CD28 вариант g) CH1- Fc-выступ PGLALA
<400> 113
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Ser Ile Tyr Pro Arg Asn Val Gln Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp His Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Glu Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Glu Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
<210> 114
<211> 448
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH (CD28 вариант f) CH1- Fc-выступ PGLALA
<400> 114
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Ser Ile Tyr Pro Gly Asn Val Gln Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Phe Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Glu Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Glu Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
<210> 115
<211> 448
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH (CD28 вариант j) CH1- Fc-выступ PGLALA
<400> 115
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Ser Ile Tyr Pro Gly Asn Val Ala Thr Arg Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Ala Asp Thr Ser Lys Asn Thr Ala Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Trp Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Glu Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Glu Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
<210> 116
<211> 448
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH (CD28 вариант e) CH1- Fc-выступ PGLALA
<400> 116
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Ser Ile Tyr Pro Gly Asn Val Gln Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Trp Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Glu Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Glu Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
<210> 117
<211> 448
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH (CD28 вариант b) CH1- Fc-выступ PGLALA
<400> 117
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Ser Ile Tyr Pro Gly Asn Val Gln Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp His Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Glu Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Glu Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
<210> 118
<211> 448
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH (CD28 вариант a) CH1- Fc-выступ PGLALA
<400> 118
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Ser Ile Tyr Pro Gly Asn Val Asn Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Trp Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Glu Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Glu Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
<210> 119
<211> 448
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH (CD28 вариант i) CH1- Fc-выступ PGLALA
<400> 119
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Ser Ile Tyr Pro Gly Asn Val Asn Thr Arg Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Ala Asp Thr Ser Lys Asn Thr Ala Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Trp Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Glu Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Glu Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
<210> 120
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL (CD28 вариант k)-CL
<400> 120
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys His Ala Ser Gln Asn Ile Tyr Val His
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ala Gln Thr Tyr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Arg Lys Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 121
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL (CD28 вариант l)-CL
<400> 121
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys His Ala Ser Gln Asn Ile Tyr Val Phe
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gly Gln Thr Tyr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Arg Lys Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 122
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL (CD28 вариант m)-CL
<400> 122
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys His Ala Ser Gln Asn Ile Tyr Val Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gly Gln Thr Tyr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Arg Lys Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 123
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL (CD28 вариант r)-CL
<400> 123
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys His Ala Ser Gln Gly Ile Tyr Val Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gly Gln Thr Tyr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Arg Lys Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 124
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL (CD28 вариант s)-CL
<400> 124
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys His Ala Ser Gln Gly Ile Ser Val Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gly Gln Thr Tyr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Arg Lys Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 125
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL (CD28 вариант t)-CL
<400> 125
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Asn Ile Tyr Val Trp
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu Tyr Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gly Gln Thr Tyr Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Arg Lys Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 126
<211> 225
<212> Белок
<213> Искусственная последовательность
<220>
<223> Fc-впадина PGLALA, HYRF
<400> 126
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly
1 5 10 15
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
20 25 30
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
35 40 45
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
50 55 60
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
65 70 75 80
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
85 90 95
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile
100 105 110
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
115 120 125
Cys Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser
130 135 140
Leu Ser Cys Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
145 150 155 160
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
165 170 175
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val
180 185 190
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
195 200 205
His Glu Ala Leu His Asn Arg Phe Thr Gln Lys Ser Leu Ser Leu Ser
210 215 220
Pro
225
<210> 127
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA CDR-H1
<400> 127
Ser Tyr Trp Met His
1 5
<210> 128
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA CDR-H2
<400> 128
Phe Ile Arg Asn Lys Ala Asn Gly Gly Thr Thr Glu Tyr Ala Ala Ser
1 5 10 15
Val Lys Gly
<210> 129
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA CDR-H3
<400> 129
Asp Arg Gly Leu Arg Phe Tyr Phe Asp Tyr
1 5 10
<210> 130
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA CDR-L1
<400> 130
Thr Leu Arg Arg Gly Ile Asn Val Gly Ala Tyr Ser Ile Tyr
1 5 10
<210> 131
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA CDR-L2
<400> 131
Tyr Lys Ser Asp Ser Asp Lys Gln Gln Gly Ser Gly Val
1 5 10
<210> 132
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA CDR-L3
<400> 132
Met Ile Trp His Ser Gly Ala Ser Ala Val
1 5 10
<210> 133
<211> 121
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA VH
<400> 133
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Val Ser Ser Tyr
20 25 30
Trp Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Phe Ile Arg Asn Lys Ala Asn Gly Gly Thr Thr Glu Tyr Ala Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Ala Arg Asp Arg Gly Leu Arg Phe Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 134
<211> 116
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA VL
<400> 134
Gln Ala Val Leu Thr Gln Pro Ala Ser Leu Ser Ala Ser Pro Gly Ala
1 5 10 15
Ser Ala Ser Leu Thr Cys Thr Leu Arg Arg Gly Ile Asn Val Gly Ala
20 25 30
Tyr Ser Ile Tyr Trp Tyr Gln Gln Lys Pro Gly Ser Pro Pro Gln Tyr
35 40 45
Leu Leu Arg Tyr Lys Ser Asp Ser Asp Lys Gln Gln Gly Ser Gly Val
50 55 60
Ser Ser Arg Phe Ser Ala Ser Lys Asp Ala Ser Ala Asn Ala Gly Ile
65 70 75 80
Leu Leu Ile Ser Gly Leu Gln Ser Glu Asp Glu Ala Asp Tyr Tyr Cys
85 90 95
Met Ile Trp His Ser Gly Ala Ser Ala Val Phe Gly Gly Gly Thr Lys
100 105 110
Leu Thr Val Leu
115
<210> 135
<211> 748
<212> Белок
<213> Искусственная последовательность
<220>
<223> His-меченный ВКД FAP человека
<400> 135
Arg Pro Ser Arg Val His Asn Ser Glu Glu Asn Thr Met Arg Ala Leu
1 5 10 15
Thr Leu Lys Asp Ile Leu Asn Gly Thr Phe Ser Tyr Lys Thr Phe Phe
20 25 30
Pro Asn Trp Ile Ser Gly Gln Glu Tyr Leu His Gln Ser Ala Asp Asn
35 40 45
Asn Ile Val Leu Tyr Asn Ile Glu Thr Gly Gln Ser Tyr Thr Ile Leu
50 55 60
Ser Asn Arg Thr Met Lys Ser Val Asn Ala Ser Asn Tyr Gly Leu Ser
65 70 75 80
Pro Asp Arg Gln Phe Val Tyr Leu Glu Ser Asp Tyr Ser Lys Leu Trp
85 90 95
Arg Tyr Ser Tyr Thr Ala Thr Tyr Tyr Ile Tyr Asp Leu Ser Asn Gly
100 105 110
Glu Phe Val Arg Gly Asn Glu Leu Pro Arg Pro Ile Gln Tyr Leu Cys
115 120 125
Trp Ser Pro Val Gly Ser Lys Leu Ala Tyr Val Tyr Gln Asn Asn Ile
130 135 140
Tyr Leu Lys Gln Arg Pro Gly Asp Pro Pro Phe Gln Ile Thr Phe Asn
145 150 155 160
Gly Arg Glu Asn Lys Ile Phe Asn Gly Ile Pro Asp Trp Val Tyr Glu
165 170 175
Glu Glu Met Leu Ala Thr Lys Tyr Ala Leu Trp Trp Ser Pro Asn Gly
180 185 190
Lys Phe Leu Ala Tyr Ala Glu Phe Asn Asp Thr Asp Ile Pro Val Ile
195 200 205
Ala Tyr Ser Tyr Tyr Gly Asp Glu Gln Tyr Pro Arg Thr Ile Asn Ile
210 215 220
Pro Tyr Pro Lys Ala Gly Ala Lys Asn Pro Val Val Arg Ile Phe Ile
225 230 235 240
Ile Asp Thr Thr Tyr Pro Ala Tyr Val Gly Pro Gln Glu Val Pro Val
245 250 255
Pro Ala Met Ile Ala Ser Ser Asp Tyr Tyr Phe Ser Trp Leu Thr Trp
260 265 270
Val Thr Asp Glu Arg Val Cys Leu Gln Trp Leu Lys Arg Val Gln Asn
275 280 285
Val Ser Val Leu Ser Ile Cys Asp Phe Arg Glu Asp Trp Gln Thr Trp
290 295 300
Asp Cys Pro Lys Thr Gln Glu His Ile Glu Glu Ser Arg Thr Gly Trp
305 310 315 320
Ala Gly Gly Phe Phe Val Ser Thr Pro Val Phe Ser Tyr Asp Ala Ile
325 330 335
Ser Tyr Tyr Lys Ile Phe Ser Asp Lys Asp Gly Tyr Lys His Ile His
340 345 350
Tyr Ile Lys Asp Thr Val Glu Asn Ala Ile Gln Ile Thr Ser Gly Lys
355 360 365
Trp Glu Ala Ile Asn Ile Phe Arg Val Thr Gln Asp Ser Leu Phe Tyr
370 375 380
Ser Ser Asn Glu Phe Glu Glu Tyr Pro Gly Arg Arg Asn Ile Tyr Arg
385 390 395 400
Ile Ser Ile Gly Ser Tyr Pro Pro Ser Lys Lys Cys Val Thr Cys His
405 410 415
Leu Arg Lys Glu Arg Cys Gln Tyr Tyr Thr Ala Ser Phe Ser Asp Tyr
420 425 430
Ala Lys Tyr Tyr Ala Leu Val Cys Tyr Gly Pro Gly Ile Pro Ile Ser
435 440 445
Thr Leu His Asp Gly Arg Thr Asp Gln Glu Ile Lys Ile Leu Glu Glu
450 455 460
Asn Lys Glu Leu Glu Asn Ala Leu Lys Asn Ile Gln Leu Pro Lys Glu
465 470 475 480
Glu Ile Lys Lys Leu Glu Val Asp Glu Ile Thr Leu Trp Tyr Lys Met
485 490 495
Ile Leu Pro Pro Gln Phe Asp Arg Ser Lys Lys Tyr Pro Leu Leu Ile
500 505 510
Gln Val Tyr Gly Gly Pro Cys Ser Gln Ser Val Arg Ser Val Phe Ala
515 520 525
Val Asn Trp Ile Ser Tyr Leu Ala Ser Lys Glu Gly Met Val Ile Ala
530 535 540
Leu Val Asp Gly Arg Gly Thr Ala Phe Gln Gly Asp Lys Leu Leu Tyr
545 550 555 560
Ala Val Tyr Arg Lys Leu Gly Val Tyr Glu Val Glu Asp Gln Ile Thr
565 570 575
Ala Val Arg Lys Phe Ile Glu Met Gly Phe Ile Asp Glu Lys Arg Ile
580 585 590
Ala Ile Trp Gly Trp Ser Tyr Gly Gly Tyr Val Ser Ser Leu Ala Leu
595 600 605
Ala Ser Gly Thr Gly Leu Phe Lys Cys Gly Ile Ala Val Ala Pro Val
610 615 620
Ser Ser Trp Glu Tyr Tyr Ala Ser Val Tyr Thr Glu Arg Phe Met Gly
625 630 635 640
Leu Pro Thr Lys Asp Asp Asn Leu Glu His Tyr Lys Asn Ser Thr Val
645 650 655
Met Ala Arg Ala Glu Tyr Phe Arg Asn Val Asp Tyr Leu Leu Ile His
660 665 670
Gly Thr Ala Asp Asp Asn Val His Phe Gln Asn Ser Ala Gln Ile Ala
675 680 685
Lys Ala Leu Val Asn Ala Gln Val Asp Phe Gln Ala Met Trp Tyr Ser
690 695 700
Asp Gln Asn His Gly Leu Ser Gly Leu Ser Thr Asn His Leu Tyr Thr
705 710 715 720
His Met Thr His Phe Leu Lys Gln Cys Phe Ser Leu Ser Asp Gly Lys
725 730 735
Lys Lys Lys Lys Lys Gly His His His His His His
740 745
<210> 136
<211> 761
<212> Белок
<213> murine
<400> 136
Met Lys Thr Trp Leu Lys Thr Val Phe Gly Val Thr Thr Leu Ala Ala
1 5 10 15
Leu Ala Leu Val Val Ile Cys Ile Val Leu Arg Pro Ser Arg Val Tyr
20 25 30
Lys Pro Glu Gly Asn Thr Lys Arg Ala Leu Thr Leu Lys Asp Ile Leu
35 40 45
Asn Gly Thr Phe Ser Tyr Lys Thr Tyr Phe Pro Asn Trp Ile Ser Glu
50 55 60
Gln Glu Tyr Leu His Gln Ser Glu Asp Asp Asn Ile Val Phe Tyr Asn
65 70 75 80
Ile Glu Thr Arg Glu Ser Tyr Ile Ile Leu Ser Asn Ser Thr Met Lys
85 90 95
Ser Val Asn Ala Thr Asp Tyr Gly Leu Ser Pro Asp Arg Gln Phe Val
100 105 110
Tyr Leu Glu Ser Asp Tyr Ser Lys Leu Trp Arg Tyr Ser Tyr Thr Ala
115 120 125
Thr Tyr Tyr Ile Tyr Asp Leu Gln Asn Gly Glu Phe Val Arg Gly Tyr
130 135 140
Glu Leu Pro Arg Pro Ile Gln Tyr Leu Cys Trp Ser Pro Val Gly Ser
145 150 155 160
Lys Leu Ala Tyr Val Tyr Gln Asn Asn Ile Tyr Leu Lys Gln Arg Pro
165 170 175
Gly Asp Pro Pro Phe Gln Ile Thr Tyr Thr Gly Arg Glu Asn Arg Ile
180 185 190
Phe Asn Gly Ile Pro Asp Trp Val Tyr Glu Glu Glu Met Leu Ala Thr
195 200 205
Lys Tyr Ala Leu Trp Trp Ser Pro Asp Gly Lys Phe Leu Ala Tyr Val
210 215 220
Glu Phe Asn Asp Ser Asp Ile Pro Ile Ile Ala Tyr Ser Tyr Tyr Gly
225 230 235 240
Asp Gly Gln Tyr Pro Arg Thr Ile Asn Ile Pro Tyr Pro Lys Ala Gly
245 250 255
Ala Lys Asn Pro Val Val Arg Val Phe Ile Val Asp Thr Thr Tyr Pro
260 265 270
His His Val Gly Pro Met Glu Val Pro Val Pro Glu Met Ile Ala Ser
275 280 285
Ser Asp Tyr Tyr Phe Ser Trp Leu Thr Trp Val Ser Ser Glu Arg Val
290 295 300
Cys Leu Gln Trp Leu Lys Arg Val Gln Asn Val Ser Val Leu Ser Ile
305 310 315 320
Cys Asp Phe Arg Glu Asp Trp His Ala Trp Glu Cys Pro Lys Asn Gln
325 330 335
Glu His Val Glu Glu Ser Arg Thr Gly Trp Ala Gly Gly Phe Phe Val
340 345 350
Ser Thr Pro Ala Phe Ser Gln Asp Ala Thr Ser Tyr Tyr Lys Ile Phe
355 360 365
Ser Asp Lys Asp Gly Tyr Lys His Ile His Tyr Ile Lys Asp Thr Val
370 375 380
Glu Asn Ala Ile Gln Ile Thr Ser Gly Lys Trp Glu Ala Ile Tyr Ile
385 390 395 400
Phe Arg Val Thr Gln Asp Ser Leu Phe Tyr Ser Ser Asn Glu Phe Glu
405 410 415
Gly Tyr Pro Gly Arg Arg Asn Ile Tyr Arg Ile Ser Ile Gly Asn Ser
420 425 430
Pro Pro Ser Lys Lys Cys Val Thr Cys His Leu Arg Lys Glu Arg Cys
435 440 445
Gln Tyr Tyr Thr Ala Ser Phe Ser Tyr Lys Ala Lys Tyr Tyr Ala Leu
450 455 460
Val Cys Tyr Gly Pro Gly Leu Pro Ile Ser Thr Leu His Asp Gly Arg
465 470 475 480
Thr Asp Gln Glu Ile Gln Val Leu Glu Glu Asn Lys Glu Leu Glu Asn
485 490 495
Ser Leu Arg Asn Ile Gln Leu Pro Lys Val Glu Ile Lys Lys Leu Lys
500 505 510
Asp Gly Gly Leu Thr Phe Trp Tyr Lys Met Ile Leu Pro Pro Gln Phe
515 520 525
Asp Arg Ser Lys Lys Tyr Pro Leu Leu Ile Gln Val Tyr Gly Gly Pro
530 535 540
Cys Ser Gln Ser Val Lys Ser Val Phe Ala Val Asn Trp Ile Thr Tyr
545 550 555 560
Leu Ala Ser Lys Glu Gly Ile Val Ile Ala Leu Val Asp Gly Arg Gly
565 570 575
Thr Ala Phe Gln Gly Asp Lys Phe Leu His Ala Val Tyr Arg Lys Leu
580 585 590
Gly Val Tyr Glu Val Glu Asp Gln Leu Thr Ala Val Arg Lys Phe Ile
595 600 605
Glu Met Gly Phe Ile Asp Glu Glu Arg Ile Ala Ile Trp Gly Trp Ser
610 615 620
Tyr Gly Gly Tyr Val Ser Ser Leu Ala Leu Ala Ser Gly Thr Gly Leu
625 630 635 640
Phe Lys Cys Gly Ile Ala Val Ala Pro Val Ser Ser Trp Glu Tyr Tyr
645 650 655
Ala Ser Ile Tyr Ser Glu Arg Phe Met Gly Leu Pro Thr Lys Asp Asp
660 665 670
Asn Leu Glu His Tyr Lys Asn Ser Thr Val Met Ala Arg Ala Glu Tyr
675 680 685
Phe Arg Asn Val Asp Tyr Leu Leu Ile His Gly Thr Ala Asp Asp Asn
690 695 700
Val His Phe Gln Asn Ser Ala Gln Ile Ala Lys Ala Leu Val Asn Ala
705 710 715 720
Gln Val Asp Phe Gln Ala Met Trp Tyr Ser Asp Gln Asn His Gly Ile
725 730 735
Ser Ser Gly Arg Ser Gln Asn His Leu Tyr Thr His Met Thr His Phe
740 745 750
Leu Lys Gln Cys Phe Ser Leu Ser Asp
755 760
<210> 137
<211> 749
<212> Белок
<213> Искусственная последовательность
<220>
<223> Мышиный эктодомен FAP +поли-lys-тег+his6-тег
<400> 137
Arg Pro Ser Arg Val Tyr Lys Pro Glu Gly Asn Thr Lys Arg Ala Leu
1 5 10 15
Thr Leu Lys Asp Ile Leu Asn Gly Thr Phe Ser Tyr Lys Thr Tyr Phe
20 25 30
Pro Asn Trp Ile Ser Glu Gln Glu Tyr Leu His Gln Ser Glu Asp Asp
35 40 45
Asn Ile Val Phe Tyr Asn Ile Glu Thr Arg Glu Ser Tyr Ile Ile Leu
50 55 60
Ser Asn Ser Thr Met Lys Ser Val Asn Ala Thr Asp Tyr Gly Leu Ser
65 70 75 80
Pro Asp Arg Gln Phe Val Tyr Leu Glu Ser Asp Tyr Ser Lys Leu Trp
85 90 95
Arg Tyr Ser Tyr Thr Ala Thr Tyr Tyr Ile Tyr Asp Leu Gln Asn Gly
100 105 110
Glu Phe Val Arg Gly Tyr Glu Leu Pro Arg Pro Ile Gln Tyr Leu Cys
115 120 125
Trp Ser Pro Val Gly Ser Lys Leu Ala Tyr Val Tyr Gln Asn Asn Ile
130 135 140
Tyr Leu Lys Gln Arg Pro Gly Asp Pro Pro Phe Gln Ile Thr Tyr Thr
145 150 155 160
Gly Arg Glu Asn Arg Ile Phe Asn Gly Ile Pro Asp Trp Val Tyr Glu
165 170 175
Glu Glu Met Leu Ala Thr Lys Tyr Ala Leu Trp Trp Ser Pro Asp Gly
180 185 190
Lys Phe Leu Ala Tyr Val Glu Phe Asn Asp Ser Asp Ile Pro Ile Ile
195 200 205
Ala Tyr Ser Tyr Tyr Gly Asp Gly Gln Tyr Pro Arg Thr Ile Asn Ile
210 215 220
Pro Tyr Pro Lys Ala Gly Ala Lys Asn Pro Val Val Arg Val Phe Ile
225 230 235 240
Val Asp Thr Thr Tyr Pro His His Val Gly Pro Met Glu Val Pro Val
245 250 255
Pro Glu Met Ile Ala Ser Ser Asp Tyr Tyr Phe Ser Trp Leu Thr Trp
260 265 270
Val Ser Ser Glu Arg Val Cys Leu Gln Trp Leu Lys Arg Val Gln Asn
275 280 285
Val Ser Val Leu Ser Ile Cys Asp Phe Arg Glu Asp Trp His Ala Trp
290 295 300
Glu Cys Pro Lys Asn Gln Glu His Val Glu Glu Ser Arg Thr Gly Trp
305 310 315 320
Ala Gly Gly Phe Phe Val Ser Thr Pro Ala Phe Ser Gln Asp Ala Thr
325 330 335
Ser Tyr Tyr Lys Ile Phe Ser Asp Lys Asp Gly Tyr Lys His Ile His
340 345 350
Tyr Ile Lys Asp Thr Val Glu Asn Ala Ile Gln Ile Thr Ser Gly Lys
355 360 365
Trp Glu Ala Ile Tyr Ile Phe Arg Val Thr Gln Asp Ser Leu Phe Tyr
370 375 380
Ser Ser Asn Glu Phe Glu Gly Tyr Pro Gly Arg Arg Asn Ile Tyr Arg
385 390 395 400
Ile Ser Ile Gly Asn Ser Pro Pro Ser Lys Lys Cys Val Thr Cys His
405 410 415
Leu Arg Lys Glu Arg Cys Gln Tyr Tyr Thr Ala Ser Phe Ser Tyr Lys
420 425 430
Ala Lys Tyr Tyr Ala Leu Val Cys Tyr Gly Pro Gly Leu Pro Ile Ser
435 440 445
Thr Leu His Asp Gly Arg Thr Asp Gln Glu Ile Gln Val Leu Glu Glu
450 455 460
Asn Lys Glu Leu Glu Asn Ser Leu Arg Asn Ile Gln Leu Pro Lys Val
465 470 475 480
Glu Ile Lys Lys Leu Lys Asp Gly Gly Leu Thr Phe Trp Tyr Lys Met
485 490 495
Ile Leu Pro Pro Gln Phe Asp Arg Ser Lys Lys Tyr Pro Leu Leu Ile
500 505 510
Gln Val Tyr Gly Gly Pro Cys Ser Gln Ser Val Lys Ser Val Phe Ala
515 520 525
Val Asn Trp Ile Thr Tyr Leu Ala Ser Lys Glu Gly Ile Val Ile Ala
530 535 540
Leu Val Asp Gly Arg Gly Thr Ala Phe Gln Gly Asp Lys Phe Leu His
545 550 555 560
Ala Val Tyr Arg Lys Leu Gly Val Tyr Glu Val Glu Asp Gln Leu Thr
565 570 575
Ala Val Arg Lys Phe Ile Glu Met Gly Phe Ile Asp Glu Glu Arg Ile
580 585 590
Ala Ile Trp Gly Trp Ser Tyr Gly Gly Tyr Val Ser Ser Leu Ala Leu
595 600 605
Ala Ser Gly Thr Gly Leu Phe Lys Cys Gly Ile Ala Val Ala Pro Val
610 615 620
Ser Ser Trp Glu Tyr Tyr Ala Ser Ile Tyr Ser Glu Arg Phe Met Gly
625 630 635 640
Leu Pro Thr Lys Asp Asp Asn Leu Glu His Tyr Lys Asn Ser Thr Val
645 650 655
Met Ala Arg Ala Glu Tyr Phe Arg Asn Val Asp Tyr Leu Leu Ile His
660 665 670
Gly Thr Ala Asp Asp Asn Val His Phe Gln Asn Ser Ala Gln Ile Ala
675 680 685
Lys Ala Leu Val Asn Ala Gln Val Asp Phe Gln Ala Met Trp Tyr Ser
690 695 700
Asp Gln Asn His Gly Ile Leu Ser Gly Arg Ser Gln Asn His Leu Tyr
705 710 715 720
Thr His Met Thr His Phe Leu Lys Gln Cys Phe Ser Leu Ser Asp Gly
725 730 735
Lys Lys Lys Lys Lys Lys Gly His His His His His His
740 745
<210> 138
<211> 748
<212> Белок
<213> Искусственная последовательность
<220>
<223> Эктодомен FAP яванского макака+поли-lys-тег+his6-тег
<400> 138
Arg Pro Pro Arg Val His Asn Ser Glu Glu Asn Thr Met Arg Ala Leu
1 5 10 15
Thr Leu Lys Asp Ile Leu Asn Gly Thr Phe Ser Tyr Lys Thr Phe Phe
20 25 30
Pro Asn Trp Ile Ser Gly Gln Glu Tyr Leu His Gln Ser Ala Asp Asn
35 40 45
Asn Ile Val Leu Tyr Asn Ile Glu Thr Gly Gln Ser Tyr Thr Ile Leu
50 55 60
Ser Asn Arg Thr Met Lys Ser Val Asn Ala Ser Asn Tyr Gly Leu Ser
65 70 75 80
Pro Asp Arg Gln Phe Val Tyr Leu Glu Ser Asp Tyr Ser Lys Leu Trp
85 90 95
Arg Tyr Ser Tyr Thr Ala Thr Tyr Tyr Ile Tyr Asp Leu Ser Asn Gly
100 105 110
Glu Phe Val Arg Gly Asn Glu Leu Pro Arg Pro Ile Gln Tyr Leu Cys
115 120 125
Trp Ser Pro Val Gly Ser Lys Leu Ala Tyr Val Tyr Gln Asn Asn Ile
130 135 140
Tyr Leu Lys Gln Arg Pro Gly Asp Pro Pro Phe Gln Ile Thr Phe Asn
145 150 155 160
Gly Arg Glu Asn Lys Ile Phe Asn Gly Ile Pro Asp Trp Val Tyr Glu
165 170 175
Glu Glu Met Leu Ala Thr Lys Tyr Ala Leu Trp Trp Ser Pro Asn Gly
180 185 190
Lys Phe Leu Ala Tyr Ala Glu Phe Asn Asp Thr Asp Ile Pro Val Ile
195 200 205
Ala Tyr Ser Tyr Tyr Gly Asp Glu Gln Tyr Pro Arg Thr Ile Asn Ile
210 215 220
Pro Tyr Pro Lys Ala Gly Ala Lys Asn Pro Phe Val Arg Ile Phe Ile
225 230 235 240
Ile Asp Thr Thr Tyr Pro Ala Tyr Val Gly Pro Gln Glu Val Pro Val
245 250 255
Pro Ala Met Ile Ala Ser Ser Asp Tyr Tyr Phe Ser Trp Leu Thr Trp
260 265 270
Val Thr Asp Glu Arg Val Cys Leu Gln Trp Leu Lys Arg Val Gln Asn
275 280 285
Val Ser Val Leu Ser Ile Cys Asp Phe Arg Glu Asp Trp Gln Thr Trp
290 295 300
Asp Cys Pro Lys Thr Gln Glu His Ile Glu Glu Ser Arg Thr Gly Trp
305 310 315 320
Ala Gly Gly Phe Phe Val Ser Thr Pro Val Phe Ser Tyr Asp Ala Ile
325 330 335
Ser Tyr Tyr Lys Ile Phe Ser Asp Lys Asp Gly Tyr Lys His Ile His
340 345 350
Tyr Ile Lys Asp Thr Val Glu Asn Ala Ile Gln Ile Thr Ser Gly Lys
355 360 365
Trp Glu Ala Ile Asn Ile Phe Arg Val Thr Gln Asp Ser Leu Phe Tyr
370 375 380
Ser Ser Asn Glu Phe Glu Asp Tyr Pro Gly Arg Arg Asn Ile Tyr Arg
385 390 395 400
Ile Ser Ile Gly Ser Tyr Pro Pro Ser Lys Lys Cys Val Thr Cys His
405 410 415
Leu Arg Lys Glu Arg Cys Gln Tyr Tyr Thr Ala Ser Phe Ser Asp Tyr
420 425 430
Ala Lys Tyr Tyr Ala Leu Val Cys Tyr Gly Pro Gly Ile Pro Ile Ser
435 440 445
Thr Leu His Asp Gly Arg Thr Asp Gln Glu Ile Lys Ile Leu Glu Glu
450 455 460
Asn Lys Glu Leu Glu Asn Ala Leu Lys Asn Ile Gln Leu Pro Lys Glu
465 470 475 480
Glu Ile Lys Lys Leu Glu Val Asp Glu Ile Thr Leu Trp Tyr Lys Met
485 490 495
Ile Leu Pro Pro Gln Phe Asp Arg Ser Lys Lys Tyr Pro Leu Leu Ile
500 505 510
Gln Val Tyr Gly Gly Pro Cys Ser Gln Ser Val Arg Ser Val Phe Ala
515 520 525
Val Asn Trp Ile Ser Tyr Leu Ala Ser Lys Glu Gly Met Val Ile Ala
530 535 540
Leu Val Asp Gly Arg Gly Thr Ala Phe Gln Gly Asp Lys Leu Leu Tyr
545 550 555 560
Ala Val Tyr Arg Lys Leu Gly Val Tyr Glu Val Glu Asp Gln Ile Thr
565 570 575
Ala Val Arg Lys Phe Ile Glu Met Gly Phe Ile Asp Glu Lys Arg Ile
580 585 590
Ala Ile Trp Gly Trp Ser Tyr Gly Gly Tyr Val Ser Ser Leu Ala Leu
595 600 605
Ala Ser Gly Thr Gly Leu Phe Lys Cys Gly Ile Ala Val Ala Pro Val
610 615 620
Ser Ser Trp Glu Tyr Tyr Ala Ser Val Tyr Thr Glu Arg Phe Met Gly
625 630 635 640
Leu Pro Thr Lys Asp Asp Asn Leu Glu His Tyr Lys Asn Ser Thr Val
645 650 655
Met Ala Arg Ala Glu Tyr Phe Arg Asn Val Asp Tyr Leu Leu Ile His
660 665 670
Gly Thr Ala Asp Asp Asn Val His Phe Gln Asn Ser Ala Gln Ile Ala
675 680 685
Lys Ala Leu Val Asn Ala Gln Val Asp Phe Gln Ala Met Trp Tyr Ser
690 695 700
Asp Gln Asn His Gly Leu Ser Gly Leu Ser Thr Asn His Leu Tyr Thr
705 710 715 720
His Met Thr His Phe Leu Lys Gln Cys Phe Ser Leu Ser Asp Gly Lys
725 730 735
Lys Lys Lys Lys Lys Gly His His His His His His
740 745
<210> 139
<211> 257
<212> Белок
<213> Homo sapiens
<400> 139
Met Ala Gln Arg Met Thr Thr Gln Leu Leu Leu Leu Leu Val Trp Val
1 5 10 15
Ala Val Val Gly Glu Ala Gln Thr Arg Ile Ala Trp Ala Arg Thr Glu
20 25 30
Leu Leu Asn Val Cys Met Asn Ala Lys His His Lys Glu Lys Pro Gly
35 40 45
Pro Glu Asp Lys Leu His Glu Gln Cys Arg Pro Trp Arg Lys Asn Ala
50 55 60
Cys Cys Ser Thr Asn Thr Ser Gln Glu Ala His Lys Asp Val Ser Tyr
65 70 75 80
Leu Tyr Arg Phe Asn Trp Asn His Cys Gly Glu Met Ala Pro Ala Cys
85 90 95
Lys Arg His Phe Ile Gln Asp Thr Cys Leu Tyr Glu Cys Ser Pro Asn
100 105 110
Leu Gly Pro Trp Ile Gln Gln Val Asp Gln Ser Trp Arg Lys Glu Arg
115 120 125
Val Leu Asn Val Pro Leu Cys Lys Glu Asp Cys Glu Gln Trp Trp Glu
130 135 140
Asp Cys Arg Thr Ser Tyr Thr Cys Lys Ser Asn Trp His Lys Gly Trp
145 150 155 160
Asn Trp Thr Ser Gly Phe Asn Lys Cys Ala Val Gly Ala Ala Cys Gln
165 170 175
Pro Phe His Phe Tyr Phe Pro Thr Pro Thr Val Leu Cys Asn Glu Ile
180 185 190
Trp Thr His Ser Tyr Lys Val Ser Asn Tyr Ser Arg Gly Ser Gly Arg
195 200 205
Cys Ile Gln Met Trp Phe Asp Pro Ala Gln Gly Asn Pro Asn Glu Glu
210 215 220
Val Ala Arg Phe Tyr Ala Ala Ala Met Ser Gly Ala Gly Pro Trp Ala
225 230 235 240
Ala Trp Pro Phe Leu Leu Ser Leu Ala Leu Met Leu Leu Trp Leu Leu
245 250 255
Ser
<210> 140
<211> 255
<212> Белок
<213> Mus musculus
<400> 140
Met Ala His Leu Met Thr Val Gln Leu Leu Leu Leu Val Met Trp Met
1 5 10 15
Ala Glu Cys Ala Gln Ser Arg Ala Thr Arg Ala Arg Thr Glu Leu Leu
20 25 30
Asn Val Cys Met Asp Ala Lys His His Lys Glu Lys Pro Gly Pro Glu
35 40 45
Asp Asn Leu His Asp Gln Cys Ser Pro Trp Lys Thr Asn Ser Cys Cys
50 55 60
Ser Thr Asn Thr Ser Gln Glu Ala His Lys Asp Ile Ser Tyr Leu Tyr
65 70 75 80
Arg Phe Asn Trp Asn His Cys Gly Thr Met Thr Ser Glu Cys Lys Arg
85 90 95
His Phe Ile Gln Asp Thr Cys Leu Tyr Glu Cys Ser Pro Asn Leu Gly
100 105 110
Pro Trp Ile Gln Gln Val Asp Gln Ser Trp Arg Lys Glu Arg Ile Leu
115 120 125
Asp Val Pro Leu Cys Lys Glu Asp Cys Gln Gln Trp Trp Glu Asp Cys
130 135 140
Gln Ser Ser Phe Thr Cys Lys Ser Asn Trp His Lys Gly Trp Asn Trp
145 150 155 160
Ser Ser Gly His Asn Glu Cys Pro Val Gly Ala Ser Cys His Pro Phe
165 170 175
Thr Phe Tyr Phe Pro Thr Ser Ala Ala Leu Cys Glu Glu Ile Trp Ser
180 185 190
His Ser Tyr Lys Leu Ser Asn Tyr Ser Arg Gly Ser Gly Arg Cys Ile
195 200 205
Gln Met Trp Phe Asp Pro Ala Gln Gly Asn Pro Asn Glu Glu Val Ala
210 215 220
Arg Phe Tyr Ala Glu Ala Met Ser Gly Ala Gly Phe His Gly Thr Trp
225 230 235 240
Pro Leu Leu Cys Ser Leu Ser Leu Val Leu Leu Trp Val Ile Ser
245 250 255
<210> 141
<211> 257
<212> Белок
<213> Яванский макак
<400> 141
Met Ala Gln Arg Met Thr Thr Gln Leu Leu Leu Leu Leu Val Trp Val
1 5 10 15
Ala Val Val Gly Glu Ala Gln Thr Arg Thr Ala Arg Ala Arg Thr Glu
20 25 30
Leu Leu Asn Val Cys Met Asn Ala Lys His His Lys Glu Lys Pro Gly
35 40 45
Pro Glu Asp Lys Leu His Glu Gln Cys Arg Pro Trp Lys Lys Asn Ala
50 55 60
Cys Cys Ser Thr Asn Thr Ser Gln Glu Ala His Lys Asp Val Ser Tyr
65 70 75 80
Leu Tyr Arg Phe Asn Trp Asn His Cys Gly Glu Met Ala Pro Ala Cys
85 90 95
Lys Arg His Phe Ile Gln Asp Thr Cys Leu Tyr Glu Cys Ser Pro Asn
100 105 110
Leu Gly Pro Trp Ile Gln Gln Val Asp Gln Ser Trp Arg Lys Glu Arg
115 120 125
Val Leu Asn Val Pro Leu Cys Lys Glu Asp Cys Glu Arg Trp Trp Glu
130 135 140
Asp Cys Arg Thr Ser Tyr Thr Cys Lys Ser Asn Trp His Lys Gly Trp
145 150 155 160
Asn Trp Thr Ser Gly Phe Asn Lys Cys Pro Val Gly Ala Ala Cys Gln
165 170 175
Pro Phe His Phe Tyr Phe Pro Thr Pro Thr Val Leu Cys Asn Glu Ile
180 185 190
Trp Thr Tyr Ser Tyr Lys Val Ser Asn Tyr Ser Arg Gly Ser Gly Arg
195 200 205
Cys Ile Gln Met Trp Phe Asp Pro Ala Gln Gly Asn Pro Asn Glu Glu
210 215 220
Val Ala Arg Phe Tyr Ala Ala Ala Met Ser Gly Ala Gly Pro Trp Ala
225 230 235 240
Ala Trp Pro Leu Leu Leu Ser Leu Ala Leu Thr Leu Leu Trp Leu Leu
245 250 255
Ser
<210> 142
<211> 2322
<212> Белок
<213> Homo sapiens
<400> 142
Met Gln Ser Gly Pro Arg Pro Pro Leu Pro Ala Pro Gly Leu Ala Leu
1 5 10 15
Ala Leu Thr Leu Thr Met Leu Ala Arg Leu Ala Ser Ala Ala Ser Phe
20 25 30
Phe Gly Glu Asn His Leu Glu Val Pro Val Ala Thr Ala Leu Thr Asp
35 40 45
Ile Asp Leu Gln Leu Gln Phe Ser Thr Ser Gln Pro Glu Ala Leu Leu
50 55 60
Leu Leu Ala Ala Gly Pro Ala Asp His Leu Leu Leu Gln Leu Tyr Ser
65 70 75 80
Gly Arg Leu Gln Val Arg Leu Val Leu Gly Gln Glu Glu Leu Arg Leu
85 90 95
Gln Thr Pro Ala Glu Thr Leu Leu Ser Asp Ser Ile Pro His Thr Val
100 105 110
Val Leu Thr Val Val Glu Gly Trp Ala Thr Leu Ser Val Asp Gly Phe
115 120 125
Leu Asn Ala Ser Ser Ala Val Pro Gly Ala Pro Leu Glu Val Pro Tyr
130 135 140
Gly Leu Phe Val Gly Gly Thr Gly Thr Leu Gly Leu Pro Tyr Leu Arg
145 150 155 160
Gly Thr Ser Arg Pro Leu Arg Gly Cys Leu His Ala Ala Thr Leu Asn
165 170 175
Gly Arg Ser Leu Leu Arg Pro Leu Thr Pro Asp Val His Glu Gly Cys
180 185 190
Ala Glu Glu Phe Ser Ala Ser Asp Asp Val Ala Leu Gly Phe Ser Gly
195 200 205
Pro His Ser Leu Ala Ala Phe Pro Ala Trp Gly Thr Gln Asp Glu Gly
210 215 220
Thr Leu Glu Phe Thr Leu Thr Thr Gln Ser Arg Gln Ala Pro Leu Ala
225 230 235 240
Phe Gln Ala Gly Gly Arg Arg Gly Asp Phe Ile Tyr Val Asp Ile Phe
245 250 255
Glu Gly His Leu Arg Ala Val Val Glu Lys Gly Gln Gly Thr Val Leu
260 265 270
Leu His Asn Ser Val Pro Val Ala Asp Gly Gln Pro His Glu Val Ser
275 280 285
Val His Ile Asn Ala His Arg Leu Glu Ile Ser Val Asp Gln Tyr Pro
290 295 300
Thr His Thr Ser Asn Arg Gly Val Leu Ser Tyr Leu Glu Pro Arg Gly
305 310 315 320
Ser Leu Leu Leu Gly Gly Leu Asp Ala Glu Ala Ser Arg His Leu Gln
325 330 335
Glu His Arg Leu Gly Leu Thr Pro Glu Ala Thr Asn Ala Ser Leu Leu
340 345 350
Gly Cys Met Glu Asp Leu Ser Val Asn Gly Gln Arg Arg Gly Leu Arg
355 360 365
Glu Ala Leu Leu Thr Arg Asn Met Ala Ala Gly Cys Arg Leu Glu Glu
370 375 380
Glu Glu Tyr Glu Asp Asp Ala Tyr Gly His Tyr Glu Ala Phe Ser Thr
385 390 395 400
Leu Ala Pro Glu Ala Trp Pro Ala Met Glu Leu Pro Glu Pro Cys Val
405 410 415
Pro Glu Pro Gly Leu Pro Pro Val Phe Ala Asn Phe Thr Gln Leu Leu
420 425 430
Thr Ile Ser Pro Leu Val Val Ala Glu Gly Gly Thr Ala Trp Leu Glu
435 440 445
Trp Arg His Val Gln Pro Thr Leu Asp Leu Met Glu Ala Glu Leu Arg
450 455 460
Lys Ser Gln Val Leu Phe Ser Val Thr Arg Gly Ala Arg His Gly Glu
465 470 475 480
Leu Glu Leu Asp Ile Pro Gly Ala Gln Ala Arg Lys Met Phe Thr Leu
485 490 495
Leu Asp Val Val Asn Arg Lys Ala Arg Phe Ile His Asp Gly Ser Glu
500 505 510
Asp Thr Ser Asp Gln Leu Val Leu Glu Val Ser Val Thr Ala Arg Val
515 520 525
Pro Met Pro Ser Cys Leu Arg Arg Gly Gln Thr Tyr Leu Leu Pro Ile
530 535 540
Gln Val Asn Pro Val Asn Asp Pro Pro His Ile Ile Phe Pro His Gly
545 550 555 560
Ser Leu Met Val Ile Leu Glu His Thr Gln Lys Pro Leu Gly Pro Glu
565 570 575
Val Phe Gln Ala Tyr Asp Pro Asp Ser Ala Cys Glu Gly Leu Thr Phe
580 585 590
Gln Val Leu Gly Thr Ser Ser Gly Leu Pro Val Glu Arg Arg Asp Gln
595 600 605
Pro Gly Glu Pro Ala Thr Glu Phe Ser Cys Arg Glu Leu Glu Ala Gly
610 615 620
Ser Leu Val Tyr Val His Arg Gly Gly Pro Ala Gln Asp Leu Thr Phe
625 630 635 640
Arg Val Ser Asp Gly Leu Gln Ala Ser Pro Pro Ala Thr Leu Lys Val
645 650 655
Val Ala Ile Arg Pro Ala Ile Gln Ile His Arg Ser Thr Gly Leu Arg
660 665 670
Leu Ala Gln Gly Ser Ala Met Pro Ile Leu Pro Ala Asn Leu Ser Val
675 680 685
Glu Thr Asn Ala Val Gly Gln Asp Val Ser Val Leu Phe Arg Val Thr
690 695 700
Gly Ala Leu Gln Phe Gly Glu Leu Gln Lys Gln Gly Ala Gly Gly Val
705 710 715 720
Glu Gly Ala Glu Trp Trp Ala Thr Gln Ala Phe His Gln Arg Asp Val
725 730 735
Glu Gln Gly Arg Val Arg Tyr Leu Ser Thr Asp Pro Gln His His Ala
740 745 750
Tyr Asp Thr Val Glu Asn Leu Ala Leu Glu Val Gln Val Gly Gln Glu
755 760 765
Ile Leu Ser Asn Leu Ser Phe Pro Val Thr Ile Gln Arg Ala Thr Val
770 775 780
Trp Met Leu Arg Leu Glu Pro Leu His Thr Gln Asn Thr Gln Gln Glu
785 790 795 800
Thr Leu Thr Thr Ala His Leu Glu Ala Thr Leu Glu Glu Ala Gly Pro
805 810 815
Ser Pro Pro Thr Phe His Tyr Glu Val Val Gln Ala Pro Arg Lys Gly
820 825 830
Asn Leu Gln Leu Gln Gly Thr Arg Leu Ser Asp Gly Gln Gly Phe Thr
835 840 845
Gln Asp Asp Ile Gln Ala Gly Arg Val Thr Tyr Gly Ala Thr Ala Arg
850 855 860
Ala Ser Glu Ala Val Glu Asp Thr Phe Arg Phe Arg Val Thr Ala Pro
865 870 875 880
Pro Tyr Phe Ser Pro Leu Tyr Thr Phe Pro Ile His Ile Gly Gly Asp
885 890 895
Pro Asp Ala Pro Val Leu Thr Asn Val Leu Leu Val Val Pro Glu Gly
900 905 910
Gly Glu Gly Val Leu Ser Ala Asp His Leu Phe Val Lys Ser Leu Asn
915 920 925
Ser Ala Ser Tyr Leu Tyr Glu Val Met Glu Arg Pro Arg His Gly Arg
930 935 940
Leu Ala Trp Arg Gly Thr Gln Asp Lys Thr Thr Met Val Thr Ser Phe
945 950 955 960
Thr Asn Glu Asp Leu Leu Arg Gly Arg Leu Val Tyr Gln His Asp Asp
965 970 975
Ser Glu Thr Thr Glu Asp Asp Ile Pro Phe Val Ala Thr Arg Gln Gly
980 985 990
Glu Ser Ser Gly Asp Met Ala Trp Glu Glu Val Arg Gly Val Phe Arg
995 1000 1005
Val Ala Ile Gln Pro Val Asn Asp His Ala Pro Val Gln Thr Ile
1010 1015 1020
Ser Arg Ile Phe His Val Ala Arg Gly Gly Arg Arg Leu Leu Thr
1025 1030 1035
Thr Asp Asp Val Ala Phe Ser Asp Ala Asp Ser Gly Phe Ala Asp
1040 1045 1050
Ala Gln Leu Val Leu Thr Arg Lys Asp Leu Leu Phe Gly Ser Ile
1055 1060 1065
Val Ala Val Asp Glu Pro Thr Arg Pro Ile Tyr Arg Phe Thr Gln
1070 1075 1080
Glu Asp Leu Arg Lys Arg Arg Val Leu Phe Val His Ser Gly Ala
1085 1090 1095
Asp Arg Gly Trp Ile Gln Leu Gln Val Ser Asp Gly Gln His Gln
1100 1105 1110
Ala Thr Ala Leu Leu Glu Val Gln Ala Ser Glu Pro Tyr Leu Arg
1115 1120 1125
Val Ala Asn Gly Ser Ser Leu Val Val Pro Gln Gly Gly Gln Gly
1130 1135 1140
Thr Ile Asp Thr Ala Val Leu His Leu Asp Thr Asn Leu Asp Ile
1145 1150 1155
Arg Ser Gly Asp Glu Val His Tyr His Val Thr Ala Gly Pro Arg
1160 1165 1170
Trp Gly Gln Leu Val Arg Ala Gly Gln Pro Ala Thr Ala Phe Ser
1175 1180 1185
Gln Gln Asp Leu Leu Asp Gly Ala Val Leu Tyr Ser His Asn Gly
1190 1195 1200
Ser Leu Ser Pro Arg Asp Thr Met Ala Phe Ser Val Glu Ala Gly
1205 1210 1215
Pro Val His Thr Asp Ala Thr Leu Gln Val Thr Ile Ala Leu Glu
1220 1225 1230
Gly Pro Leu Ala Pro Leu Lys Leu Val Arg His Lys Lys Ile Tyr
1235 1240 1245
Val Phe Gln Gly Glu Ala Ala Glu Ile Arg Arg Asp Gln Leu Glu
1250 1255 1260
Ala Ala Gln Glu Ala Val Pro Pro Ala Asp Ile Val Phe Ser Val
1265 1270 1275
Lys Ser Pro Pro Ser Ala Gly Tyr Leu Val Met Val Ser Arg Gly
1280 1285 1290
Ala Leu Ala Asp Glu Pro Pro Ser Leu Asp Pro Val Gln Ser Phe
1295 1300 1305
Ser Gln Glu Ala Val Asp Thr Gly Arg Val Leu Tyr Leu His Ser
1310 1315 1320
Arg Pro Glu Ala Trp Ser Asp Ala Phe Ser Leu Asp Val Ala Ser
1325 1330 1335
Gly Leu Gly Ala Pro Leu Glu Gly Val Leu Val Glu Leu Glu Val
1340 1345 1350
Leu Pro Ala Ala Ile Pro Leu Glu Ala Gln Asn Phe Ser Val Pro
1355 1360 1365
Glu Gly Gly Ser Leu Thr Leu Ala Pro Pro Leu Leu Arg Val Ser
1370 1375 1380
Gly Pro Tyr Phe Pro Thr Leu Leu Gly Leu Ser Leu Gln Val Leu
1385 1390 1395
Glu Pro Pro Gln His Gly Ala Leu Gln Lys Glu Asp Gly Pro Gln
1400 1405 1410
Ala Arg Thr Leu Ser Ala Phe Ser Trp Arg Met Val Glu Glu Gln
1415 1420 1425
Leu Ile Arg Tyr Val His Asp Gly Ser Glu Thr Leu Thr Asp Ser
1430 1435 1440
Phe Val Leu Met Ala Asn Ala Ser Glu Met Asp Arg Gln Ser His
1445 1450 1455
Pro Val Ala Phe Thr Val Thr Val Leu Pro Val Asn Asp Gln Pro
1460 1465 1470
Pro Ile Leu Thr Thr Asn Thr Gly Leu Gln Met Trp Glu Gly Ala
1475 1480 1485
Thr Ala Pro Ile Pro Ala Glu Ala Leu Arg Ser Thr Asp Gly Asp
1490 1495 1500
Ser Gly Ser Glu Asp Leu Val Tyr Thr Ile Glu Gln Pro Ser Asn
1505 1510 1515
Gly Arg Val Val Leu Arg Gly Ala Pro Gly Thr Glu Val Arg Ser
1520 1525 1530
Phe Thr Gln Ala Gln Leu Asp Gly Gly Leu Val Leu Phe Ser His
1535 1540 1545
Arg Gly Thr Leu Asp Gly Gly Phe Arg Phe Arg Leu Ser Asp Gly
1550 1555 1560
Glu His Thr Ser Pro Gly His Phe Phe Arg Val Thr Ala Gln Lys
1565 1570 1575
Gln Val Leu Leu Ser Leu Lys Gly Ser Gln Thr Leu Thr Val Cys
1580 1585 1590
Pro Gly Ser Val Gln Pro Leu Ser Ser Gln Thr Leu Arg Ala Ser
1595 1600 1605
Ser Ser Ala Gly Thr Asp Pro Gln Leu Leu Leu Tyr Arg Val Val
1610 1615 1620
Arg Gly Pro Gln Leu Gly Arg Leu Phe His Ala Gln Gln Asp Ser
1625 1630 1635
Thr Gly Glu Ala Leu Val Asn Phe Thr Gln Ala Glu Val Tyr Ala
1640 1645 1650
Gly Asn Ile Leu Tyr Glu His Glu Met Pro Pro Glu Pro Phe Trp
1655 1660 1665
Glu Ala His Asp Thr Leu Glu Leu Gln Leu Ser Ser Pro Pro Ala
1670 1675 1680
Arg Asp Val Ala Ala Thr Leu Ala Val Ala Val Ser Phe Glu Ala
1685 1690 1695
Ala Cys Pro Gln Arg Pro Ser His Leu Trp Lys Asn Lys Gly Leu
1700 1705 1710
Trp Val Pro Glu Gly Gln Arg Ala Arg Ile Thr Val Ala Ala Leu
1715 1720 1725
Asp Ala Ser Asn Leu Leu Ala Ser Val Pro Ser Pro Gln Arg Ser
1730 1735 1740
Glu His Asp Val Leu Phe Gln Val Thr Gln Phe Pro Ser Arg Gly
1745 1750 1755
Gln Leu Leu Val Ser Glu Glu Pro Leu His Ala Gly Gln Pro His
1760 1765 1770
Phe Leu Gln Ser Gln Leu Ala Ala Gly Gln Leu Val Tyr Ala His
1775 1780 1785
Gly Gly Gly Gly Thr Gln Gln Asp Gly Phe His Phe Arg Ala His
1790 1795 1800
Leu Gln Gly Pro Ala Gly Ala Ser Val Ala Gly Pro Gln Thr Ser
1805 1810 1815
Glu Ala Phe Ala Ile Thr Val Arg Asp Val Asn Glu Arg Pro Pro
1820 1825 1830
Gln Pro Gln Ala Ser Val Pro Leu Arg Leu Thr Arg Gly Ser Arg
1835 1840 1845
Ala Pro Ile Ser Arg Ala Gln Leu Ser Val Val Asp Pro Asp Ser
1850 1855 1860
Ala Pro Gly Glu Ile Glu Tyr Glu Val Gln Arg Ala Pro His Asn
1865 1870 1875
Gly Phe Leu Ser Leu Val Gly Gly Gly Leu Gly Pro Val Thr Arg
1880 1885 1890
Phe Thr Gln Ala Asp Val Asp Ser Gly Arg Leu Ala Phe Val Ala
1895 1900 1905
Asn Gly Ser Ser Val Ala Gly Ile Phe Gln Leu Ser Met Ser Asp
1910 1915 1920
Gly Ala Ser Pro Pro Leu Pro Met Ser Leu Ala Val Asp Ile Leu
1925 1930 1935
Pro Ser Ala Ile Glu Val Gln Leu Arg Ala Pro Leu Glu Val Pro
1940 1945 1950
Gln Ala Leu Gly Arg Ser Ser Leu Ser Gln Gln Gln Leu Arg Val
1955 1960 1965
Val Ser Asp Arg Glu Glu Pro Glu Ala Ala Tyr Arg Leu Ile Gln
1970 1975 1980
Gly Pro Gln Tyr Gly His Leu Leu Val Gly Gly Arg Pro Thr Ser
1985 1990 1995
Ala Phe Ser Gln Phe Gln Ile Asp Gln Gly Glu Val Val Phe Ala
2000 2005 2010
Phe Thr Asn Phe Ser Ser Ser His Asp His Phe Arg Val Leu Ala
2015 2020 2025
Leu Ala Arg Gly Val Asn Ala Ser Ala Val Val Asn Val Thr Val
2030 2035 2040
Arg Ala Leu Leu His Val Trp Ala Gly Gly Pro Trp Pro Gln Gly
2045 2050 2055
Ala Thr Leu Arg Leu Asp Pro Thr Val Leu Asp Ala Gly Glu Leu
2060 2065 2070
Ala Asn Arg Thr Gly Ser Val Pro Arg Phe Arg Leu Leu Glu Gly
2075 2080 2085
Pro Arg His Gly Arg Val Val Arg Val Pro Arg Ala Arg Thr Glu
2090 2095 2100
Pro Gly Gly Ser Gln Leu Val Glu Gln Phe Thr Gln Gln Asp Leu
2105 2110 2115
Glu Asp Gly Arg Leu Gly Leu Glu Val Gly Arg Pro Glu Gly Arg
2120 2125 2130
Ala Pro Gly Pro Ala Gly Asp Ser Leu Thr Leu Glu Leu Trp Ala
2135 2140 2145
Gln Gly Val Pro Pro Ala Val Ala Ser Leu Asp Phe Ala Thr Glu
2150 2155 2160
Pro Tyr Asn Ala Ala Arg Pro Tyr Ser Val Ala Leu Leu Ser Val
2165 2170 2175
Pro Glu Ala Ala Arg Thr Glu Ala Gly Lys Pro Glu Ser Ser Thr
2180 2185 2190
Pro Thr Gly Glu Pro Gly Pro Met Ala Ser Ser Pro Glu Pro Ala
2195 2200 2205
Val Ala Lys Gly Gly Phe Leu Ser Phe Leu Glu Ala Asn Met Phe
2210 2215 2220
Ser Val Ile Ile Pro Met Cys Leu Val Leu Leu Leu Leu Ala Leu
2225 2230 2235
Ile Leu Pro Leu Leu Phe Tyr Leu Arg Lys Arg Asn Lys Thr Gly
2240 2245 2250
Lys His Asp Val Gln Val Leu Thr Ala Lys Pro Arg Asn Gly Leu
2255 2260 2265
Ala Gly Asp Thr Glu Thr Phe Arg Lys Val Glu Pro Gly Gln Ala
2270 2275 2280
Ile Pro Leu Thr Ala Val Pro Gly Gln Gly Pro Pro Pro Gly Gly
2285 2290 2295
Gln Pro Asp Pro Glu Leu Leu Gln Phe Cys Arg Thr Pro Asn Pro
2300 2305 2310
Ala Leu Lys Asn Gly Gln Tyr Trp Val
2315 2320
<210> 143
<211> 1210
<212> Белок
<213> Homo sapiens
<400> 143
Met Arg Pro Ser Gly Thr Ala Gly Ala Ala Leu Leu Ala Leu Leu Ala
1 5 10 15
Ala Leu Cys Pro Ala Ser Arg Ala Leu Glu Glu Lys Lys Val Cys Gln
20 25 30
Gly Thr Ser Asn Lys Leu Thr Gln Leu Gly Thr Phe Glu Asp His Phe
35 40 45
Leu Ser Leu Gln Arg Met Phe Asn Asn Cys Glu Val Val Leu Gly Asn
50 55 60
Leu Glu Ile Thr Tyr Val Gln Arg Asn Tyr Asp Leu Ser Phe Leu Lys
65 70 75 80
Thr Ile Gln Glu Val Ala Gly Tyr Val Leu Ile Ala Leu Asn Thr Val
85 90 95
Glu Arg Ile Pro Leu Glu Asn Leu Gln Ile Ile Arg Gly Asn Met Tyr
100 105 110
Tyr Glu Asn Ser Tyr Ala Leu Ala Val Leu Ser Asn Tyr Asp Ala Asn
115 120 125
Lys Thr Gly Leu Lys Glu Leu Pro Met Arg Asn Leu Gln Glu Ile Leu
130 135 140
His Gly Ala Val Arg Phe Ser Asn Asn Pro Ala Leu Cys Asn Val Glu
145 150 155 160
Ser Ile Gln Trp Arg Asp Ile Val Ser Ser Asp Phe Leu Ser Asn Met
165 170 175
Ser Met Asp Phe Gln Asn His Leu Gly Ser Cys Gln Lys Cys Asp Pro
180 185 190
Ser Cys Pro Asn Gly Ser Cys Trp Gly Ala Gly Glu Glu Asn Cys Gln
195 200 205
Lys Leu Thr Lys Ile Ile Cys Ala Gln Gln Cys Ser Gly Arg Cys Arg
210 215 220
Gly Lys Ser Pro Ser Asp Cys Cys His Asn Gln Cys Ala Ala Gly Cys
225 230 235 240
Thr Gly Pro Arg Glu Ser Asp Cys Leu Val Cys Arg Lys Phe Arg Asp
245 250 255
Glu Ala Thr Cys Lys Asp Thr Cys Pro Pro Leu Met Leu Tyr Asn Pro
260 265 270
Thr Thr Tyr Gln Met Asp Val Asn Pro Glu Gly Lys Tyr Ser Phe Gly
275 280 285
Ala Thr Cys Val Lys Lys Cys Pro Arg Asn Tyr Val Val Thr Asp His
290 295 300
Gly Ser Cys Val Arg Ala Cys Gly Ala Asp Ser Tyr Glu Met Glu Glu
305 310 315 320
Asp Gly Val Arg Lys Cys Lys Lys Cys Glu Gly Pro Cys Arg Lys Val
325 330 335
Cys Asn Gly Ile Gly Ile Gly Glu Phe Lys Asp Ser Leu Ser Ile Asn
340 345 350
Ala Thr Asn Ile Lys His Phe Lys Asn Cys Thr Ser Ile Ser Gly Asp
355 360 365
Leu His Ile Leu Pro Val Ala Phe Arg Gly Asp Ser Phe Thr His Thr
370 375 380
Pro Pro Leu Asp Pro Gln Glu Leu Asp Ile Leu Lys Thr Val Lys Glu
385 390 395 400
Ile Thr Gly Phe Leu Leu Ile Gln Ala Trp Pro Glu Asn Arg Thr Asp
405 410 415
Leu His Ala Phe Glu Asn Leu Glu Ile Ile Arg Gly Arg Thr Lys Gln
420 425 430
His Gly Gln Phe Ser Leu Ala Val Val Ser Leu Asn Ile Thr Ser Leu
435 440 445
Gly Leu Arg Ser Leu Lys Glu Ile Ser Asp Gly Asp Val Ile Ile Ser
450 455 460
Gly Asn Lys Asn Leu Cys Tyr Ala Asn Thr Ile Asn Trp Lys Lys Leu
465 470 475 480
Phe Gly Thr Ser Gly Gln Lys Thr Lys Ile Ile Ser Asn Arg Gly Glu
485 490 495
Asn Ser Cys Lys Ala Thr Gly Gln Val Cys His Ala Leu Cys Ser Pro
500 505 510
Glu Gly Cys Trp Gly Pro Glu Pro Arg Asp Cys Val Ser Cys Arg Asn
515 520 525
Val Ser Arg Gly Arg Glu Cys Val Asp Lys Cys Asn Leu Leu Glu Gly
530 535 540
Glu Pro Arg Glu Phe Val Glu Asn Ser Glu Cys Ile Gln Cys His Pro
545 550 555 560
Glu Cys Leu Pro Gln Ala Met Asn Ile Thr Cys Thr Gly Arg Gly Pro
565 570 575
Asp Asn Cys Ile Gln Cys Ala His Tyr Ile Asp Gly Pro His Cys Val
580 585 590
Lys Thr Cys Pro Ala Gly Val Met Gly Glu Asn Asn Thr Leu Val Trp
595 600 605
Lys Tyr Ala Asp Ala Gly His Val Cys His Leu Cys His Pro Asn Cys
610 615 620
Thr Tyr Gly Cys Thr Gly Pro Gly Leu Glu Gly Cys Pro Thr Asn Gly
625 630 635 640
Pro Lys Ile Pro Ser Ile Ala Thr Gly Met Val Gly Ala Leu Leu Leu
645 650 655
Leu Leu Val Val Ala Leu Gly Ile Gly Leu Phe Met Arg Arg Arg His
660 665 670
Ile Val Arg Lys Arg Thr Leu Arg Arg Leu Leu Gln Glu Arg Glu Leu
675 680 685
Val Glu Pro Leu Thr Pro Ser Gly Glu Ala Pro Asn Gln Ala Leu Leu
690 695 700
Arg Ile Leu Lys Glu Thr Glu Phe Lys Lys Ile Lys Val Leu Gly Ser
705 710 715 720
Gly Ala Phe Gly Thr Val Tyr Lys Gly Leu Trp Ile Pro Glu Gly Glu
725 730 735
Lys Val Lys Ile Pro Val Ala Ile Lys Glu Leu Arg Glu Ala Thr Ser
740 745 750
Pro Lys Ala Asn Lys Glu Ile Leu Asp Glu Ala Tyr Val Met Ala Ser
755 760 765
Val Asp Asn Pro His Val Cys Arg Leu Leu Gly Ile Cys Leu Thr Ser
770 775 780
Thr Val Gln Leu Ile Thr Gln Leu Met Pro Phe Gly Cys Leu Leu Asp
785 790 795 800
Tyr Val Arg Glu His Lys Asp Asn Ile Gly Ser Gln Tyr Leu Leu Asn
805 810 815
Trp Cys Val Gln Ile Ala Lys Gly Met Asn Tyr Leu Glu Asp Arg Arg
820 825 830
Leu Val His Arg Asp Leu Ala Ala Arg Asn Val Leu Val Lys Thr Pro
835 840 845
Gln His Val Lys Ile Thr Asp Phe Gly Leu Ala Lys Leu Leu Gly Ala
850 855 860
Glu Glu Lys Glu Tyr His Ala Glu Gly Gly Lys Val Pro Ile Lys Trp
865 870 875 880
Met Ala Leu Glu Ser Ile Leu His Arg Ile Tyr Thr His Gln Ser Asp
885 890 895
Val Trp Ser Tyr Gly Val Thr Val Trp Glu Leu Met Thr Phe Gly Ser
900 905 910
Lys Pro Tyr Asp Gly Ile Pro Ala Ser Glu Ile Ser Ser Ile Leu Glu
915 920 925
Lys Gly Glu Arg Leu Pro Gln Pro Pro Ile Cys Thr Ile Asp Val Tyr
930 935 940
Met Ile Met Val Lys Cys Trp Met Ile Asp Ala Asp Ser Arg Pro Lys
945 950 955 960
Phe Arg Glu Leu Ile Ile Glu Phe Ser Lys Met Ala Arg Asp Pro Gln
965 970 975
Arg Tyr Leu Val Ile Gln Gly Asp Glu Arg Met His Leu Pro Ser Pro
980 985 990
Thr Asp Ser Asn Phe Tyr Arg Ala Leu Met Asp Glu Glu Asp Met Asp
995 1000 1005
Asp Val Val Asp Ala Asp Glu Tyr Leu Ile Pro Gln Gln Gly Phe
1010 1015 1020
Phe Ser Ser Pro Ser Thr Ser Arg Thr Pro Leu Leu Ser Ser Leu
1025 1030 1035
Ser Ala Thr Ser Asn Asn Ser Thr Val Ala Cys Ile Asp Arg Asn
1040 1045 1050
Gly Leu Gln Ser Cys Pro Ile Lys Glu Asp Ser Phe Leu Gln Arg
1055 1060 1065
Tyr Ser Ser Asp Pro Thr Gly Ala Leu Thr Glu Asp Ser Ile Asp
1070 1075 1080
Asp Thr Phe Leu Pro Val Pro Glu Tyr Ile Asn Gln Ser Val Pro
1085 1090 1095
Lys Arg Pro Ala Gly Ser Val Gln Asn Pro Val Tyr His Asn Gln
1100 1105 1110
Pro Leu Asn Pro Ala Pro Ser Arg Asp Pro His Tyr Gln Asp Pro
1115 1120 1125
His Ser Thr Ala Val Gly Asn Pro Glu Tyr Leu Asn Thr Val Gln
1130 1135 1140
Pro Thr Cys Val Asn Ser Thr Phe Asp Ser Pro Ala His Trp Ala
1145 1150 1155
Gln Lys Gly Ser His Gln Ile Ser Leu Asp Asn Pro Asp Tyr Gln
1160 1165 1170
Gln Asp Phe Phe Pro Lys Glu Ala Lys Pro Asn Gly Ile Phe Lys
1175 1180 1185
Gly Ser Thr Ala Glu Asn Ala Glu Tyr Leu Arg Val Ala Pro Gln
1190 1195 1200
Ser Ser Glu Phe Ile Gly Ala
1205 1210
<210> 144
<211> 1255
<212> Белок
<213> Homo sapiens
<400> 144
Met Glu Leu Ala Ala Leu Cys Arg Trp Gly Leu Leu Leu Ala Leu Leu
1 5 10 15
Pro Pro Gly Ala Ala Ser Thr Gln Val Cys Thr Gly Thr Asp Met Lys
20 25 30
Leu Arg Leu Pro Ala Ser Pro Glu Thr His Leu Asp Met Leu Arg His
35 40 45
Leu Tyr Gln Gly Cys Gln Val Val Gln Gly Asn Leu Glu Leu Thr Tyr
50 55 60
Leu Pro Thr Asn Ala Ser Leu Ser Phe Leu Gln Asp Ile Gln Glu Val
65 70 75 80
Gln Gly Tyr Val Leu Ile Ala His Asn Gln Val Arg Gln Val Pro Leu
85 90 95
Gln Arg Leu Arg Ile Val Arg Gly Thr Gln Leu Phe Glu Asp Asn Tyr
100 105 110
Ala Leu Ala Val Leu Asp Asn Gly Asp Pro Leu Asn Asn Thr Thr Pro
115 120 125
Val Thr Gly Ala Ser Pro Gly Gly Leu Arg Glu Leu Gln Leu Arg Ser
130 135 140
Leu Thr Glu Ile Leu Lys Gly Gly Val Leu Ile Gln Arg Asn Pro Gln
145 150 155 160
Leu Cys Tyr Gln Asp Thr Ile Leu Trp Lys Asp Ile Phe His Lys Asn
165 170 175
Asn Gln Leu Ala Leu Thr Leu Ile Asp Thr Asn Arg Ser Arg Ala Cys
180 185 190
His Pro Cys Ser Pro Met Cys Lys Gly Ser Arg Cys Trp Gly Glu Ser
195 200 205
Ser Glu Asp Cys Gln Ser Leu Thr Arg Thr Val Cys Ala Gly Gly Cys
210 215 220
Ala Arg Cys Lys Gly Pro Leu Pro Thr Asp Cys Cys His Glu Gln Cys
225 230 235 240
Ala Ala Gly Cys Thr Gly Pro Lys His Ser Asp Cys Leu Ala Cys Leu
245 250 255
His Phe Asn His Ser Gly Ile Cys Glu Leu His Cys Pro Ala Leu Val
260 265 270
Thr Tyr Asn Thr Asp Thr Phe Glu Ser Met Pro Asn Pro Glu Gly Arg
275 280 285
Tyr Thr Phe Gly Ala Ser Cys Val Thr Ala Cys Pro Tyr Asn Tyr Leu
290 295 300
Ser Thr Asp Val Gly Ser Cys Thr Leu Val Cys Pro Leu His Asn Gln
305 310 315 320
Glu Val Thr Ala Glu Asp Gly Thr Gln Arg Cys Glu Lys Cys Ser Lys
325 330 335
Pro Cys Ala Arg Val Cys Tyr Gly Leu Gly Met Glu His Leu Arg Glu
340 345 350
Val Arg Ala Val Thr Ser Ala Asn Ile Gln Glu Phe Ala Gly Cys Lys
355 360 365
Lys Ile Phe Gly Ser Leu Ala Phe Leu Pro Glu Ser Phe Asp Gly Asp
370 375 380
Pro Ala Ser Asn Thr Ala Pro Leu Gln Pro Glu Gln Leu Gln Val Phe
385 390 395 400
Glu Thr Leu Glu Glu Ile Thr Gly Tyr Leu Tyr Ile Ser Ala Trp Pro
405 410 415
Asp Ser Leu Pro Asp Leu Ser Val Phe Gln Asn Leu Gln Val Ile Arg
420 425 430
Gly Arg Ile Leu His Asn Gly Ala Tyr Ser Leu Thr Leu Gln Gly Leu
435 440 445
Gly Ile Ser Trp Leu Gly Leu Arg Ser Leu Arg Glu Leu Gly Ser Gly
450 455 460
Leu Ala Leu Ile His His Asn Thr His Leu Cys Phe Val His Thr Val
465 470 475 480
Pro Trp Asp Gln Leu Phe Arg Asn Pro His Gln Ala Leu Leu His Thr
485 490 495
Ala Asn Arg Pro Glu Asp Glu Cys Val Gly Glu Gly Leu Ala Cys His
500 505 510
Gln Leu Cys Ala Arg Gly His Cys Trp Gly Pro Gly Pro Thr Gln Cys
515 520 525
Val Asn Cys Ser Gln Phe Leu Arg Gly Gln Glu Cys Val Glu Glu Cys
530 535 540
Arg Val Leu Gln Gly Leu Pro Arg Glu Tyr Val Asn Ala Arg His Cys
545 550 555 560
Leu Pro Cys His Pro Glu Cys Gln Pro Gln Asn Gly Ser Val Thr Cys
565 570 575
Phe Gly Pro Glu Ala Asp Gln Cys Val Ala Cys Ala His Tyr Lys Asp
580 585 590
Pro Pro Phe Cys Val Ala Arg Cys Pro Ser Gly Val Lys Pro Asp Leu
595 600 605
Ser Tyr Met Pro Ile Trp Lys Phe Pro Asp Glu Glu Gly Ala Cys Gln
610 615 620
Pro Cys Pro Ile Asn Cys Thr His Ser Cys Val Asp Leu Asp Asp Lys
625 630 635 640
Gly Cys Pro Ala Glu Gln Arg Ala Ser Pro Leu Thr Ser Ile Ile Ser
645 650 655
Ala Val Val Gly Ile Leu Leu Val Val Val Leu Gly Val Val Phe Gly
660 665 670
Ile Leu Ile Lys Arg Arg Gln Gln Lys Ile Arg Lys Tyr Thr Met Arg
675 680 685
Arg Leu Leu Gln Glu Thr Glu Leu Val Glu Pro Leu Thr Pro Ser Gly
690 695 700
Ala Met Pro Asn Gln Ala Gln Met Arg Ile Leu Lys Glu Thr Glu Leu
705 710 715 720
Arg Lys Val Lys Val Leu Gly Ser Gly Ala Phe Gly Thr Val Tyr Lys
725 730 735
Gly Ile Trp Ile Pro Asp Gly Glu Asn Val Lys Ile Pro Val Ala Ile
740 745 750
Lys Val Leu Arg Glu Asn Thr Ser Pro Lys Ala Asn Lys Glu Ile Leu
755 760 765
Asp Glu Ala Tyr Val Met Ala Gly Val Gly Ser Pro Tyr Val Ser Arg
770 775 780
Leu Leu Gly Ile Cys Leu Thr Ser Thr Val Gln Leu Val Thr Gln Leu
785 790 795 800
Met Pro Tyr Gly Cys Leu Leu Asp His Val Arg Glu Asn Arg Gly Arg
805 810 815
Leu Gly Ser Gln Asp Leu Leu Asn Trp Cys Met Gln Ile Ala Lys Gly
820 825 830
Met Ser Tyr Leu Glu Asp Val Arg Leu Val His Arg Asp Leu Ala Ala
835 840 845
Arg Asn Val Leu Val Lys Ser Pro Asn His Val Lys Ile Thr Asp Phe
850 855 860
Gly Leu Ala Arg Leu Leu Asp Ile Asp Glu Thr Glu Tyr His Ala Asp
865 870 875 880
Gly Gly Lys Val Pro Ile Lys Trp Met Ala Leu Glu Ser Ile Leu Arg
885 890 895
Arg Arg Phe Thr His Gln Ser Asp Val Trp Ser Tyr Gly Val Thr Val
900 905 910
Trp Glu Leu Met Thr Phe Gly Ala Lys Pro Tyr Asp Gly Ile Pro Ala
915 920 925
Arg Glu Ile Pro Asp Leu Leu Glu Lys Gly Glu Arg Leu Pro Gln Pro
930 935 940
Pro Ile Cys Thr Ile Asp Val Tyr Met Ile Met Val Lys Cys Trp Met
945 950 955 960
Ile Asp Ser Glu Cys Arg Pro Arg Phe Arg Glu Leu Val Ser Glu Phe
965 970 975
Ser Arg Met Ala Arg Asp Pro Gln Arg Phe Val Val Ile Gln Asn Glu
980 985 990
Asp Leu Gly Pro Ala Ser Pro Leu Asp Ser Thr Phe Tyr Arg Ser Leu
995 1000 1005
Leu Glu Asp Asp Asp Met Gly Asp Leu Val Asp Ala Glu Glu Tyr
1010 1015 1020
Leu Val Pro Gln Gln Gly Phe Phe Cys Pro Asp Pro Ala Pro Gly
1025 1030 1035
Ala Gly Gly Met Val His His Arg His Arg Ser Ser Ser Thr Arg
1040 1045 1050
Ser Gly Gly Gly Asp Leu Thr Leu Gly Leu Glu Pro Ser Glu Glu
1055 1060 1065
Glu Ala Pro Arg Ser Pro Leu Ala Pro Ser Glu Gly Ala Gly Ser
1070 1075 1080
Asp Val Phe Asp Gly Asp Leu Gly Met Gly Ala Ala Lys Gly Leu
1085 1090 1095
Gln Ser Leu Pro Thr His Asp Pro Ser Pro Leu Gln Arg Tyr Ser
1100 1105 1110
Glu Asp Pro Thr Val Pro Leu Pro Ser Glu Thr Asp Gly Tyr Val
1115 1120 1125
Ala Pro Leu Thr Cys Ser Pro Gln Pro Glu Tyr Val Asn Gln Pro
1130 1135 1140
Asp Val Arg Pro Gln Pro Pro Ser Pro Arg Glu Gly Pro Leu Pro
1145 1150 1155
Ala Ala Arg Pro Ala Gly Ala Thr Leu Glu Arg Pro Lys Thr Leu
1160 1165 1170
Ser Pro Gly Lys Asn Gly Val Val Lys Asp Val Phe Ala Phe Gly
1175 1180 1185
Gly Ala Val Glu Asn Pro Glu Tyr Leu Thr Pro Gln Gly Gly Ala
1190 1195 1200
Ala Pro Gln Pro His Pro Pro Pro Ala Phe Ser Pro Ala Phe Asp
1205 1210 1215
Asn Leu Tyr Tyr Trp Asp Gln Asp Pro Pro Glu Arg Gly Ala Pro
1220 1225 1230
Pro Ser Thr Phe Lys Gly Thr Pro Thr Ala Glu Asn Pro Glu Tyr
1235 1240 1245
Leu Gly Leu Asp Val Pro Val
1250 1255
<210> 145
<211> 645
<212> Белок
<213> Homo sapiens
<400> 145
Met Pro Ile Trp Lys Phe Pro Asp Glu Glu Gly Ala Cys Gln Pro Cys
1 5 10 15
Pro Ile Asn Cys Thr His Ser Cys Val Asp Leu Asp Asp Lys Gly Cys
20 25 30
Pro Ala Glu Gln Arg Ala Ser Pro Leu Thr Ser Ile Ile Ser Ala Val
35 40 45
Val Gly Ile Leu Leu Val Val Val Leu Gly Val Val Phe Gly Ile Leu
50 55 60
Ile Lys Arg Arg Gln Gln Lys Ile Arg Lys Tyr Thr Met Arg Arg Leu
65 70 75 80
Leu Gln Glu Thr Glu Leu Val Glu Pro Leu Thr Pro Ser Gly Ala Met
85 90 95
Pro Asn Gln Ala Gln Met Arg Ile Leu Lys Glu Thr Glu Leu Arg Lys
100 105 110
Val Lys Val Leu Gly Ser Gly Ala Phe Gly Thr Val Tyr Lys Gly Ile
115 120 125
Trp Ile Pro Asp Gly Glu Asn Val Lys Ile Pro Val Ala Ile Lys Val
130 135 140
Leu Arg Glu Asn Thr Ser Pro Lys Ala Asn Lys Glu Ile Leu Asp Glu
145 150 155 160
Ala Tyr Val Met Ala Gly Val Gly Ser Pro Tyr Val Ser Arg Leu Leu
165 170 175
Gly Ile Cys Leu Thr Ser Thr Val Gln Leu Val Thr Gln Leu Met Pro
180 185 190
Tyr Gly Cys Leu Leu Asp His Val Arg Glu Asn Arg Gly Arg Leu Gly
195 200 205
Ser Gln Asp Leu Leu Asn Trp Cys Met Gln Ile Ala Lys Gly Met Ser
210 215 220
Tyr Leu Glu Asp Val Arg Leu Val His Arg Asp Leu Ala Ala Arg Asn
225 230 235 240
Val Leu Val Lys Ser Pro Asn His Val Lys Ile Thr Asp Phe Gly Leu
245 250 255
Ala Arg Leu Leu Asp Ile Asp Glu Thr Glu Tyr His Ala Asp Gly Gly
260 265 270
Lys Val Pro Ile Lys Trp Met Ala Leu Glu Ser Ile Leu Arg Arg Arg
275 280 285
Phe Thr His Gln Ser Asp Val Trp Ser Tyr Gly Val Thr Val Trp Glu
290 295 300
Leu Met Thr Phe Gly Ala Lys Pro Tyr Asp Gly Ile Pro Ala Arg Glu
305 310 315 320
Ile Pro Asp Leu Leu Glu Lys Gly Glu Arg Leu Pro Gln Pro Pro Ile
325 330 335
Cys Thr Ile Asp Val Tyr Met Ile Met Val Lys Cys Trp Met Ile Asp
340 345 350
Ser Glu Cys Arg Pro Arg Phe Arg Glu Leu Val Ser Glu Phe Ser Arg
355 360 365
Met Ala Arg Asp Pro Gln Arg Phe Val Val Ile Gln Asn Glu Asp Leu
370 375 380
Gly Pro Ala Ser Pro Leu Asp Ser Thr Phe Tyr Arg Ser Leu Leu Glu
385 390 395 400
Asp Asp Asp Met Gly Asp Leu Val Asp Ala Glu Glu Tyr Leu Val Pro
405 410 415
Gln Gln Gly Phe Phe Cys Pro Asp Pro Ala Pro Gly Ala Gly Gly Met
420 425 430
Val His His Arg His Arg Ser Ser Ser Thr Arg Ser Gly Gly Gly Asp
435 440 445
Leu Thr Leu Gly Leu Glu Pro Ser Glu Glu Glu Ala Pro Arg Ser Pro
450 455 460
Leu Ala Pro Ser Glu Gly Ala Gly Ser Asp Val Phe Asp Gly Asp Leu
465 470 475 480
Gly Met Gly Ala Ala Lys Gly Leu Gln Ser Leu Pro Thr His Asp Pro
485 490 495
Ser Pro Leu Gln Arg Tyr Ser Glu Asp Pro Thr Val Pro Leu Pro Ser
500 505 510
Glu Thr Asp Gly Tyr Val Ala Pro Leu Thr Cys Ser Pro Gln Pro Glu
515 520 525
Tyr Val Asn Gln Pro Asp Val Arg Pro Gln Pro Pro Ser Pro Arg Glu
530 535 540
Gly Pro Leu Pro Ala Ala Arg Pro Ala Gly Ala Thr Leu Glu Arg Pro
545 550 555 560
Lys Thr Leu Ser Pro Gly Lys Asn Gly Val Val Lys Asp Val Phe Ala
565 570 575
Phe Gly Gly Ala Val Glu Asn Pro Glu Tyr Leu Thr Pro Gln Gly Gly
580 585 590
Ala Ala Pro Gln Pro His Pro Pro Pro Ala Phe Ser Pro Ala Phe Asp
595 600 605
Asn Leu Tyr Tyr Trp Asp Gln Asp Pro Pro Glu Arg Gly Ala Pro Pro
610 615 620
Ser Thr Phe Lys Gly Thr Pro Thr Ala Glu Asn Pro Glu Tyr Leu Gly
625 630 635 640
Leu Asp Val Pro Val
645
<210> 146
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер G4S
<400> 146
Gly Gly Gly Gly Ser
1 5
<210> 147
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер (G4S)2
<400> 147
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
1 5 10
<210> 148
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер (SG4)2
<400> 148
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
1 5 10
<210> 149
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер G4(SG4)2
<400> 149
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
1 5 10
<210> 150
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер
<400> 150
Gly Ser Pro Gly Ser Ser Ser Ser Gly Ser
1 5 10
<210> 151
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер (G4S)3
<400> 151
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
1 5 10 15
<210> 152
<211> 20
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер (G4S)4
<400> 152
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
1 5 10 15
Gly Gly Gly Ser
20
<210> 153
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер
<400> 153
Gly Ser Gly Ser Gly Ser Gly Ser
1 5
<210> 154
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер
<400> 154
Gly Ser Gly Ser Gly Asn Gly Ser
1 5
<210> 155
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер
<400> 155
Gly Gly Ser Gly Ser Gly Ser Gly
1 5
<210> 156
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер
<400> 156
Gly Gly Ser Gly Ser Gly
1 5
<210> 157
<211> 4
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер
<400> 157
Gly Gly Ser Gly
1
<210> 158
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер
<400> 158
Gly Gly Ser Gly Asn Gly Ser Gly
1 5
<210> 159
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер
<400> 159
Gly Gly Asn Gly Ser Gly Ser Gly
1 5
<210> 160
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер
<400> 160
Gly Gly Asn Gly Ser Gly
1 5
<210> 161
<211> 215
<212> Белок
<213> Искусственная последовательность
<220>
<223> Легкая цепь CEA 2F1" (CEA TCB)
<400> 161
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Ala Ala Val Gly Thr Tyr
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ser Ala Ser Tyr Arg Lys Arg Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys His Gln Tyr Tyr Thr Tyr Pro Leu
85 90 95
Phe Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Arg Thr Val Ala
100 105 110
Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser
115 120 125
Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu
130 135 140
Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser
145 150 155 160
Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu
165 170 175
Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val
180 185 190
Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys
195 200 205
Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 162
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD3 CH2527 с гуманизированной легкой цепью (кросс-fab, VL-CH1)
(CEA TCB)
<400> 162
Gln Ala Val Val Thr Gln Glu Pro Ser Leu Thr Val Ser Pro Gly Gly
1 5 10 15
Thr Val Thr Leu Thr Cys Gly Ser Ser Thr Gly Ala Val Thr Thr Ser
20 25 30
Asn Tyr Ala Asn Trp Val Gln Glu Lys Pro Gly Gln Ala Phe Arg Gly
35 40 45
Leu Ile Gly Gly Thr Asn Lys Arg Ala Pro Gly Thr Pro Ala Arg Phe
50 55 60
Ser Gly Ser Leu Leu Gly Gly Lys Ala Ala Leu Thr Leu Ser Gly Ala
65 70 75 80
Gln Pro Glu Asp Glu Ala Glu Tyr Tyr Cys Ala Leu Trp Tyr Ser Asn
85 90 95
Leu Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Ser Ser Ala
100 105 110
Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser
115 120 125
Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe
130 135 140
Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly
145 150 155 160
Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu
165 170 175
Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr
180 185 190
Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys
195 200 205
Val Glu Pro Lys Ser Cys
210
<210> 163
<211> 692
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA CH1A1A 98/99 - гуманизированная CD3 CH2527 (кросс-fab
VH-Ck)-Fc(выступ)
P329GLALA (CEA TCB)
<400> 163
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Glu Phe
20 25 30
Gly Met Asn Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Asn Thr Lys Thr Gly Glu Ala Thr Tyr Val Glu Glu Phe
50 55 60
Lys Gly Arg Val Thr Phe Thr Thr Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Trp Asp Phe Ala Tyr Tyr Val Glu Ala Met Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
210 215 220
Asp Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Val Gln Leu Leu
225 230 235 240
Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser
245 250 255
Cys Ala Ala Ser Gly Phe Thr Phe Ser Thr Tyr Ala Met Asn Trp Val
260 265 270
Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ser Arg Ile Arg Ser
275 280 285
Lys Tyr Asn Asn Tyr Ala Thr Tyr Tyr Ala Asp Ser Val Lys Gly Arg
290 295 300
Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr Leu Tyr Leu Gln Met
305 310 315 320
Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Val Arg His
325 330 335
Gly Asn Phe Gly Asn Ser Tyr Val Ser Trp Phe Ala Tyr Trp Gly Gln
340 345 350
Gly Thr Leu Val Thr Val Ser Ser Ala Ser Val Ala Ala Pro Ser Val
355 360 365
Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser
370 375 380
Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln
385 390 395 400
Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val
405 410 415
Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu
420 425 430
Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu
435 440 445
Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg
450 455 460
Gly Glu Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
465 470 475 480
Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
485 490 495
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
500 505 510
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
515 520 525
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
530 535 540
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
545 550 555 560
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly
565 570 575
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
580 585 590
Pro Gln Val Tyr Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn
595 600 605
Gln Val Ser Leu Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
610 615 620
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
625 630 635 640
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
645 650 655
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
660 665 670
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
675 680 685
Ser Leu Ser Pro
690
<210> 164
<211> 449
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA CH1A1A 98/99 (VH-CH1)-Fc(впадина) P329GLALA (CEA TCB)
<400> 164
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Glu Phe
20 25 30
Gly Met Asn Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Asn Thr Lys Thr Gly Glu Ala Thr Tyr Val Glu Glu Phe
50 55 60
Lys Gly Arg Val Thr Phe Thr Thr Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Trp Asp Phe Ala Tyr Tyr Val Glu Ala Met Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
210 215 220
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly
225 230 235 240
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
245 250 255
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
260 265 270
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
275 280 285
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
290 295 300
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
305 310 315 320
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile
325 330 335
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
340 345 350
Cys Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser
355 360 365
Leu Ser Cys Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
370 375 380
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
385 390 395 400
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val
405 410 415
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
420 425 430
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
435 440 445
Pro
<210> 165
<211> 232
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD3 VH-CL (CEACAM5 TCB)
<400> 165
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Thr Tyr
20 25 30
Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Arg Ile Arg Ser Lys Tyr Asn Asn Tyr Ala Thr Tyr Tyr Ala Asp
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Val Arg His Gly Asn Phe Gly Asn Ser Tyr Val Ser Trp Phe
100 105 110
Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Val
115 120 125
Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys
130 135 140
Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg
145 150 155 160
Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn
165 170 175
Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser
180 185 190
Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys
195 200 205
Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr
210 215 220
Lys Ser Phe Asn Arg Gly Glu Cys
225 230
<210> 166
<211> 449
<212> Белок
<213> Искусственная последовательность
<220>
<223> гуманизированная CEA VH-CH1(EE)- Fc антигена (впадина, P329G
LALA) (CEACAM5 TCB)
<400> 166
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Phe Asn Ile Lys Asp Thr
20 25 30
Tyr Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Arg Ile Asp Pro Ala Asn Gly Asn Ser Lys Tyr Val Pro Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Pro Phe Gly Tyr Tyr Val Ser Asp Tyr Ala Met Ala Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Glu Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Glu Lys Val Glu Pro Lys Ser Cys
210 215 220
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly
225 230 235 240
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
245 250 255
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
260 265 270
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
275 280 285
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
290 295 300
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
305 310 315 320
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile
325 330 335
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
340 345 350
Cys Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser
355 360 365
Leu Ser Cys Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
370 375 380
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
385 390 395 400
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val
405 410 415
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
420 425 430
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
435 440 445
Pro
<210> 167
<211> 674
<212> Белок
<213> Искусственная последовательность
<220>
<223> гуманизированная CEA VH-CH1(EE)-CD3 VL-CH1-Fc (выступ, P329G
LALA)
(CEACAM5 TCB)
<400> 167
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Phe Asn Ile Lys Asp Thr
20 25 30
Tyr Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Arg Ile Asp Pro Ala Asn Gly Asn Ser Lys Tyr Val Pro Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Pro Phe Gly Tyr Tyr Val Ser Asp Tyr Ala Met Ala Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Glu Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Glu Lys Val Glu Pro Lys Ser Cys
210 215 220
Asp Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ala Val Val Thr
225 230 235 240
Gln Glu Pro Ser Leu Thr Val Ser Pro Gly Gly Thr Val Thr Leu Thr
245 250 255
Cys Gly Ser Ser Thr Gly Ala Val Thr Thr Ser Asn Tyr Ala Asn Trp
260 265 270
Val Gln Glu Lys Pro Gly Gln Ala Phe Arg Gly Leu Ile Gly Gly Thr
275 280 285
Asn Lys Arg Ala Pro Gly Thr Pro Ala Arg Phe Ser Gly Ser Leu Leu
290 295 300
Gly Gly Lys Ala Ala Leu Thr Leu Ser Gly Ala Gln Pro Glu Asp Glu
305 310 315 320
Ala Glu Tyr Tyr Cys Ala Leu Trp Tyr Ser Asn Leu Trp Val Phe Gly
325 330 335
Gly Gly Thr Lys Leu Thr Val Leu Ser Ser Ala Ser Thr Lys Gly Pro
340 345 350
Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr
355 360 365
Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr
370 375 380
Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro
385 390 395 400
Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr
405 410 415
Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn
420 425 430
His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser
435 440 445
Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala
450 455 460
Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu
465 470 475 480
Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser
485 490 495
His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu
500 505 510
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr
515 520 525
Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn
530 535 540
Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro
545 550 555 560
Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln
565 570 575
Val Tyr Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val
580 585 590
Ser Leu Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val
595 600 605
Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro
610 615 620
Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr
625 630 635 640
Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val
645 650 655
Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu
660 665 670
Ser Pro
<210> 168
<211> 218
<212> Белок
<213> Искусственная последовательность
<220>
<223> гуманизированная CEA VL-CL(RK) (CEACAM5 TCB)
<400> 168
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Gly Glu Ser Val Asp Ile Phe
20 25 30
Gly Val Gly Phe Leu His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro
35 40 45
Arg Leu Leu Ile Tyr Arg Ala Ser Asn Arg Ala Thr Gly Ile Pro Ala
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
65 70 75 80
Ser Leu Glu Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Thr Asn
85 90 95
Glu Asp Pro Tyr Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Arg
100 105 110
Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Arg Lys
115 120 125
Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr
130 135 140
Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser
145 150 155 160
Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr
165 170 175
Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys
180 185 190
His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro
195 200 205
Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 169
<211> 420
<212> Белок
<213> Искусственная последовательность
<220>
<223> антиген на основе CEACAM5 Hu N(A2-B2)A-avi-His
<400> 169
Gln Leu Thr Thr Glu Ser Met Pro Phe Asn Val Ala Glu Gly Lys Glu
1 5 10 15
Val Leu Leu Leu Val His Asn Leu Pro Gln Gln Leu Phe Gly Tyr Ser
20 25 30
Trp Tyr Lys Gly Glu Arg Val Asp Gly Asn Arg Gln Ile Val Gly Tyr
35 40 45
Ala Ile Gly Thr Gln Gln Ala Thr Pro Gly Pro Ala Asn Ser Gly Arg
50 55 60
Glu Thr Ile Tyr Pro Asn Ala Ser Leu Leu Ile Gln Asn Val Thr Gln
65 70 75 80
Asn Asp Thr Gly Phe Tyr Thr Leu Gln Val Ile Lys Ser Asp Leu Val
85 90 95
Asn Glu Glu Ala Thr Gly Gln Phe His Val Tyr Pro Glu Leu Pro Lys
100 105 110
Pro Phe Ile Thr Ser Asn Asn Ser Asn Pro Val Glu Asp Glu Asp Ala
115 120 125
Val Ala Leu Thr Cys Glu Pro Glu Ile Gln Asn Thr Thr Tyr Leu Trp
130 135 140
Trp Val Asn Asn Gln Ser Leu Pro Val Ser Pro Arg Leu Gln Leu Ser
145 150 155 160
Asn Asp Asn Arg Thr Leu Thr Leu Leu Ser Val Thr Arg Asn Asp Val
165 170 175
Gly Pro Tyr Glu Cys Gly Ile Gln Asn Lys Leu Ser Val Asp His Ser
180 185 190
Asp Pro Val Ile Leu Asn Val Leu Tyr Gly Pro Asp Asp Pro Thr Ile
195 200 205
Ser Pro Ser Tyr Thr Tyr Tyr Arg Pro Gly Val Asn Leu Ser Leu Ser
210 215 220
Cys His Ala Ala Ser Asn Pro Pro Ala Gln Tyr Ser Trp Leu Ile Asp
225 230 235 240
Gly Asn Ile Gln Gln His Thr Gln Glu Leu Phe Ile Ser Asn Ile Thr
245 250 255
Glu Lys Asn Ser Gly Leu Tyr Thr Cys Gln Ala Asn Asn Ser Ala Ser
260 265 270
Gly His Ser Arg Thr Thr Val Lys Thr Ile Thr Val Ser Ala Leu Ser
275 280 285
Pro Val Val Ala Lys Pro Gln Ile Lys Ala Ser Lys Thr Thr Val Thr
290 295 300
Gly Asp Lys Asp Ser Val Asn Leu Thr Cys Ser Thr Asn Asp Thr Gly
305 310 315 320
Ile Ser Ile Arg Trp Phe Phe Lys Asn Gln Ser Leu Pro Ser Ser Glu
325 330 335
Arg Met Lys Leu Ser Gln Gly Asn Ile Thr Leu Ser Ile Asn Pro Val
340 345 350
Lys Arg Glu Asp Ala Gly Thr Tyr Trp Cys Glu Val Phe Asn Pro Ile
355 360 365
Ser Lys Asn Gln Ser Asp Pro Ile Met Leu Asn Val Asn Tyr Asn Ala
370 375 380
Leu Pro Gln Glu Asn Leu Ile Asn Val Asp Gly Ser Gly Leu Asn Asp
385 390 395 400
Ile Phe Glu Ala Gln Lys Ile Glu Trp His Glu Ala Arg Ala His His
405 410 415
His His His His
420
<210> 170
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (A5B7)- CDR-H1
<400> 170
Asp Tyr Tyr Met Asn
1 5
<210> 171
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (A5B7)- CDR-H2
<400> 171
Phe Ile Gly Asn Lys Ala Asn Gly Tyr Thr Thr Glu Tyr Ser Ala Ser
1 5 10 15
Val Lys Gly
<210> 172
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (A5B7)- CDR-H3
<400> 172
Asp Arg Gly Leu Arg Phe Tyr Phe Asp Tyr
1 5 10
<210> 173
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (A5B7)- CDR-L1
<400> 173
Arg Ala Ser Ser Ser Val Thr Tyr Ile His
1 5 10
<210> 174
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (A5B7)- CDR-L2
<400> 174
Ala Thr Ser Asn Leu Ala Ser
1 5
<210> 175
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (A5B7)- CDR-L3
<400> 175
Gln His Trp Ser Ser Lys Pro Pro Thr
1 5
<210> 176
<211> 215
<212> Белок
<213> Искусственная последовательность
<220>
<223> IgG1 Fc-выступ PGLALA
<400> 176
Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
1 5 10 15
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
20 25 30
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
35 40 45
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
50 55 60
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
65 70 75 80
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
85 90 95
Ala Leu Gly Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
100 105 110
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Cys Arg Asp Glu Leu
115 120 125
Thr Lys Asn Gln Val Ser Leu Trp Cys Leu Val Lys Gly Phe Tyr Pro
130 135 140
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
145 150 155 160
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
165 170 175
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
180 185 190
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
195 200 205
Lys Ser Leu Ser Leu Ser Pro
210 215
<210> 177
<211> 215
<212> Белок
<213> Искусственная последовательность
<220>
<223> IgG1 Fc-впадина PGLALA
<400> 177
Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
1 5 10 15
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
20 25 30
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
35 40 45
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
50 55 60
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
65 70 75 80
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
85 90 95
Ala Leu Gly Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
100 105 110
Pro Arg Glu Pro Gln Val Cys Thr Leu Pro Pro Ser Arg Asp Glu Leu
115 120 125
Thr Lys Asn Gln Val Ser Leu Ser Cys Ala Val Lys Gly Phe Tyr Pro
130 135 140
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
145 150 155 160
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
165 170 175
Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
180 185 190
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
195 200 205
Lys Ser Leu Ser Leu Ser Pro
210 215
<210> 178
<211> 121
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (A5B7) VH (родительская)
<400> 178
Glu Val Lys Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Thr Ser Gly Phe Thr Phe Thr Asp Tyr
20 25 30
Tyr Met Asn Trp Val Arg Gln Pro Pro Gly Lys Ala Leu Glu Trp Leu
35 40 45
Gly Phe Ile Gly Asn Lys Ala Asn Gly Tyr Thr Thr Glu Tyr Ser Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Lys Ser Gln Ser Ile
65 70 75 80
Leu Tyr Leu Gln Met Asn Thr Leu Arg Ala Glu Asp Ser Ala Thr Tyr
85 90 95
Tyr Cys Thr Arg Asp Arg Gly Leu Arg Phe Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Leu Thr Val Ser Ser
115 120
<210> 179
<211> 106
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (A5B7) VL (родительская)
<400> 179
Gln Thr Val Leu Ser Gln Ser Pro Ala Ile Leu Ser Ala Ser Pro Gly
1 5 10 15
Glu Lys Val Thr Met Thr Cys Arg Ala Ser Ser Ser Val Thr Tyr Ile
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Ser Ser Pro Lys Ser Trp Ile Tyr
35 40 45
Ala Thr Ser Asn Leu Ala Ser Gly Val Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Ser Tyr Ser Leu Thr Ile Ser Arg Val Glu Ala Glu
65 70 75 80
Asp Ala Ala Thr Tyr Tyr Cys Gln His Trp Ser Ser Lys Pro Pro Thr
85 90 95
Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 180
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (A5H1EL1D)- CDR-H1
<400> 180
Gly Phe Thr Phe Thr Asp Tyr Tyr Met Asn
1 5 10
<210> 181
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (A5H1EL1D)- CDR-H2
<400> 181
Phe Ile Gly Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala Ser
1 5 10 15
Val Lys Gly
<210> 182
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (A5H1EL1D)- CDR-H3
<400> 182
Asp Arg Gly Leu Arg Phe Tyr Phe Asp Tyr
1 5 10
<210> 183
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (A5H1EL1D)- CDR-L1
<400> 183
Arg Ala Ser Ser Ser Val Thr Tyr Ile His
1 5 10
<210> 184
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (A5H1EL1D)- CDR-L2
<400> 184
Ala Thr Ser Asn Leu Ala Ser
1 5
<210> 185
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (A5H1EL1D)- CDR-L3
<400> 185
Gln His Trp Ser Ser Lys Pro Pro Thr
1 5
<210> 186
<211> 121
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (A5H1EL1D) VH (3-23A5-1E)
<400> 186
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Thr Asp Tyr
20 25 30
Tyr Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Phe Ile Gly Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Lys Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr
85 90 95
Tyr Cys Thr Arg Asp Arg Gly Leu Arg Phe Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 187
<211> 106
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (A5H1EL1D) VL (A5-L1D)
<400> 187
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Ser Ser Val Thr Tyr Ile
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Ser Trp Ile Tyr
35 40 45
Ala Thr Ser Asn Leu Ala Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu
65 70 75 80
Asp Phe Ala Val Tyr Tyr Cys Gln His Trp Ser Ser Lys Pro Pro Thr
85 90 95
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 188
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (A5H1EL1D aff. mat.) CDR-H1 консенсусная
<220>
<221> ВАРИАНТ
<222> (3)..(3)
<223> X is Thr или Tyr
<220>
<221> ВАРИАНТ
<222> (5)..(5)
<223> X is Thr или Ser
<220>
<221> ВАРИАНТ
<222> (8)..(8)
<223> X is Tyr или Ala или Glu
<400> 188
Gly Phe Xaa Phe Xaa Asp Tyr Xaa Met Asn
1 5 10
<210> 189
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (A5H1EL1D aff. mat.) CDR-H2 консенсусная
<220>
<221> ВАРИАНТ
<222> (1)..(1)
<223> X is Phe или Val
<220>
<221> ВАРИАНТ
<222> (3)..(3)
<223> X is Gly или Ser
<400> 189
Xaa Ile Xaa Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala Ser
1 5 10 15
Val Lys Gly
<210> 190
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (A5H1EL1D aff. mat.) CDR-H3 консенсусная
<220>
<221> ВАРИАНТ
<222> (4)..(4)
<223> X is Leu или Ile
<220>
<221> ВАРИАНТ
<222> (7)..(7)
<223> X is Tyr или Gly или Gln или Ser
<400> 190
Asp Arg Gly Xaa Arg Phe Xaa Phe Asp Tyr
1 5 10
<210> 191
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (A5H1EL1D aff. mat.) CDR-L1 консенсусная
<220>
<221> ВАРИАНТ
<222> (1)..(1)
<223> X is Arg или His
<400> 191
Xaa Ala Ser Ser Ser Val Thr Tyr Ile His
1 5 10
<210> 192
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (A5H1EL1D aff. mat.) CDR-L2 консенсусная
<400> 192
Ala Thr Ser Asn Leu Ala Ser
1 5
<210> 193
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (A5H1EL1D aff. mat.) CDR-L3 консенсусная
<220>
<221> ВАРИАНТ
<222> (6)..(6)
<223> X is Lys или Val или Gln или Ile
<220>
<221> ВАРИАНТ
<222> (7)..(7)
<223> X is Pro или Ser
<400> 193
Gln His Trp Ser Ser Xaa Xaa Pro Thr
1 5
<210> 194
<211> 121
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P006.038) VH
<400> 194
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Thr Asp Tyr
20 25 30
Tyr Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Phe Ile Gly Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Lys Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr
85 90 95
Tyr Cys Thr Arg Asp Arg Gly Ile Arg Phe Gly Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 195
<211> 106
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P006.038) VL
<400> 195
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Ser Ser Val Thr Tyr Ile
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Ser Trp Ile Tyr
35 40 45
Ala Thr Ser Asn Leu Ala Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu
65 70 75 80
Asp Phe Ala Val Tyr Tyr Cys Gln His Trp Ser Ser Val Pro Pro Thr
85 90 95
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 196
<211> 121
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.097) VH
<400> 196
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Thr Asp Tyr
20 25 30
Tyr Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Phe Ile Gly Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Lys Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr
85 90 95
Tyr Cys Thr Arg Asp Arg Gly Leu Arg Phe Ser Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 197
<211> 106
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.097) VL
<400> 197
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Ser Ser Val Thr Tyr Ile
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Ser Trp Ile Tyr
35 40 45
Ala Thr Ser Asn Leu Ala Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu
65 70 75 80
Asp Phe Ala Val Tyr Tyr Cys Gln His Trp Ser Ser Gln Pro Pro Thr
85 90 95
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 198
<211> 121
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.103) VH
<400> 198
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Thr Asp Tyr
20 25 30
Tyr Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Phe Ile Gly Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Lys Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr
85 90 95
Tyr Cys Thr Arg Asp Arg Gly Ile Arg Phe Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 199
<211> 106
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.103) VL
<400> 199
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Ser Ser Val Thr Tyr Ile
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Ser Trp Ile Tyr
35 40 45
Ala Thr Ser Asn Leu Ala Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu
65 70 75 80
Asp Phe Ala Val Tyr Tyr Cys Gln His Trp Ser Ser Ile Ser Pro Thr
85 90 95
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 200
<211> 121
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P002.139) VH
<400> 200
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Tyr Phe Thr Asp Tyr
20 25 30
Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Val Ile Ser Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Lys Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr
85 90 95
Tyr Cys Thr Arg Asp Arg Gly Leu Arg Phe Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 201
<211> 106
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P002.139) VL
<400> 201
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys His Ala Ser Ser Ser Val Thr Tyr Ile
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Ser Trp Ile Tyr
35 40 45
Ala Thr Ser Asn Leu Ala Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu
65 70 75 80
Asp Phe Ala Val Tyr Tyr Cys Gln His Trp Ser Ser Lys Pro Pro Thr
85 90 95
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 202
<211> 121
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P001.177) VH
<400> 202
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Tyr Phe Thr Asp Tyr
20 25 30
Tyr Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Phe Ile Ser Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Lys Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr
85 90 95
Tyr Cys Thr Arg Asp Arg Gly Leu Arg Phe Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 203
<211> 106
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P001.177) VL
<400> 203
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Ser Ser Val Thr Tyr Ile
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Ser Trp Ile Tyr
35 40 45
Ala Thr Ser Asn Leu Ala Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu
65 70 75 80
Asp Phe Ala Val Tyr Tyr Cys Gln His Trp Ser Ser Lys Pro Pro Thr
85 90 95
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 204
<211> 121
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.102) VH
<400> 204
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Thr Asp Tyr
20 25 30
Tyr Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Phe Ile Gly Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Lys Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr
85 90 95
Tyr Cys Thr Arg Asp Arg Gly Ile Arg Phe Gln Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 205
<211> 106
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.102) VL
<400> 205
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Ser Ser Val Thr Tyr Ile
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Ser Trp Ile Tyr
35 40 45
Ala Thr Ser Asn Leu Ala Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu
65 70 75 80
Asp Phe Ala Val Tyr Tyr Cys Gln His Trp Ser Ser Lys Ser Pro Thr
85 90 95
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 206
<211> 121
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.102 комбо1) VH
<400> 206
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Tyr Phe Thr Asp Tyr
20 25 30
Tyr Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Val Ile Ser Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Lys Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr
85 90 95
Tyr Cys Thr Arg Asp Arg Gly Ile Arg Phe Gln Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 207
<211> 106
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.102 комбо1) VL
<400> 207
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Ser Ser Val Thr Tyr Ile
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Ser Trp Ile Tyr
35 40 45
Ala Thr Ser Asn Leu Ala Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu
65 70 75 80
Asp Phe Ala Val Tyr Tyr Cys Gln His Trp Ser Ser Lys Ser Pro Thr
85 90 95
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 208
<211> 121
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.102 комбо2) VH
<400> 208
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Tyr Phe Ser Asp Tyr
20 25 30
Tyr Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Val Ile Ser Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Lys Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr
85 90 95
Tyr Cys Thr Arg Asp Arg Gly Ile Arg Phe Gln Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 209
<211> 106
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.102 комбо2) VL
<400> 209
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Ser Ser Val Thr Tyr Ile
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Ser Trp Ile Tyr
35 40 45
Ala Thr Ser Asn Leu Ala Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu
65 70 75 80
Asp Phe Ala Val Tyr Tyr Cys Gln His Trp Ser Ser Lys Ser Pro Thr
85 90 95
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 210
<211> 121
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.103 комбо1) VH
<400> 210
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Thr Asp Tyr
20 25 30
Tyr Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Phe Ile Gly Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Lys Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr
85 90 95
Tyr Cys Thr Arg Asp Arg Gly Ile Arg Phe Ser Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 211
<211> 106
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.103 комбо1) VL
<400> 211
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Ser Ser Val Thr Tyr Ile
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Ser Trp Ile Tyr
35 40 45
Ala Thr Ser Asn Leu Ala Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu
65 70 75 80
Asp Phe Ala Val Tyr Tyr Cys Gln His Trp Ser Ser Ile Ser Pro Thr
85 90 95
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 212
<211> 121
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.103 комбо2) VH
<400> 212
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Tyr Phe Thr Asp Tyr
20 25 30
Tyr Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Val Ile Ser Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Lys Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr
85 90 95
Tyr Cys Thr Arg Asp Arg Gly Ile Arg Phe Ser Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 213
<211> 106
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.103 комбо2) VL
<400> 213
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Ser Ser Val Thr Tyr Ile
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Ser Trp Ile Tyr
35 40 45
Ala Thr Ser Asn Leu Ala Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu
65 70 75 80
Asp Phe Ala Val Tyr Tyr Cys Gln His Trp Ser Ser Ile Ser Pro Thr
85 90 95
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 214
<211> 121
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P006.038 комбо1) VH
<400> 214
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Tyr Phe Thr Asp Tyr
20 25 30
Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Val Ile Ser Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Lys Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr
85 90 95
Tyr Cys Thr Arg Asp Arg Gly Ile Arg Phe Gly Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 215
<211> 106
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P006.038 комбо1) VL
<400> 215
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Ser Ser Val Thr Tyr Ile
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Ser Trp Ile Tyr
35 40 45
Ala Thr Ser Asn Leu Ala Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu
65 70 75 80
Asp Phe Ala Val Tyr Tyr Cys Gln His Trp Ser Ser Val Pro Pro Thr
85 90 95
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 216
<211> 121
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P006.038 комбо2) VH
<400> 216
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Tyr
20 25 30
Glu Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Phe Ile Ser Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Lys Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr
85 90 95
Tyr Cys Thr Arg Asp Arg Gly Ile Arg Phe Gly Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 217
<211> 106
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P006.038 комбо2) VL
<400> 217
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Ser Ser Val Thr Tyr Ile
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Ser Trp Ile Tyr
35 40 45
Ala Thr Ser Asn Leu Ala Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu
65 70 75 80
Asp Phe Ala Val Tyr Tyr Cys Gln His Trp Ser Ser Val Pro Pro Thr
85 90 95
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 218
<211> 98
<212> Белок
<213> Искусственная последовательность
<220>
<223> IGHV3-23-02
<400> 218
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Gly Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys
<210> 219
<211> 100
<212> Белок
<213> Искусственная последовательность
<220>
<223> IGHV3-15*01
<400> 219
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asn Ala
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Arg Ile Lys Ser Lys Thr Asp Gly Gly Thr Thr Asp Tyr Ala Ala
50 55 60
Pro Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Thr Thr
100
<210> 220
<211> 121
<212> Белок
<213> Искусственная последовательность
<220>
<223> 3-23A5-1
<400> 220
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Thr Asp Tyr
20 25 30
Tyr Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Phe Ile Gly Asn Lys Ala Asn Gly Tyr Thr Thr Glu Tyr Ser Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Ala Arg Asp Arg Gly Leu Arg Phe Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 221
<211> 121
<212> Белок
<213> Искусственная последовательность
<220>
<223> 3-23A5-2
<400> 221
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Thr Asp Tyr
20 25 30
Tyr Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Phe Ile Gly Asn Lys Ala Asn Gly Tyr Thr Thr Tyr Tyr Gly Asp
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Ala Arg Asp Arg Gly Leu Arg Phe Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 222
<211> 119
<212> Белок
<213> Искусственная последовательность
<220>
<223> 3-23A5-3
<400> 222
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Thr Asp Tyr
20 25 30
Tyr Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Phe Ile Gly Asn Lys Gly Tyr Thr Thr Glu Tyr Ser Ala Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Arg Gly Leu Arg Phe Tyr Phe Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Thr Val Thr Val Ser Ser
115
<210> 223
<211> 121
<212> Белок
<213> Искусственная последовательность
<220>
<223> 3-23A5-4
<400> 223
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Thr Asp Tyr
20 25 30
Tyr Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Phe Ile Gly Asn Lys Ala Asn Gly Tyr Thr Thr Glu Tyr Ser Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Ala Arg Asp Arg Gly Leu Arg Phe Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 224
<211> 121
<212> Белок
<213> Искусственная последовательность
<220>
<223> 3-23A5-1A (все обратные мутации)
<400> 224
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Thr Asp Tyr
20 25 30
Tyr Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Phe Ile Gly Asn Lys Ala Asn Gly Tyr Thr Thr Glu Tyr Ser Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Lys Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr
85 90 95
Tyr Cys Thr Arg Asp Arg Gly Leu Arg Phe Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 225
<211> 121
<212> Белок
<213> Искусственная последовательность
<220>
<223> 3-23A5-1C (A93T)
<400> 225
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Thr Asp Tyr
20 25 30
Tyr Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Phe Ile Gly Asn Lys Ala Asn Gly Tyr Thr Thr Glu Tyr Ser Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Thr Arg Asp Arg Gly Leu Arg Phe Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 226
<211> 121
<212> Белок
<213> Искусственная последовательность
<220>
<223> 3-23A5-1D (K73)
<400> 226
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Thr Asp Tyr
20 25 30
Tyr Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Phe Ile Gly Asn Lys Ala Asn Gly Tyr Thr Thr Glu Tyr Ser Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Lys Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Ala Arg Asp Arg Gly Leu Arg Phe Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 227
<211> 121
<212> Белок
<213> Искусственная последовательность
<220>
<223> 3-15A5-1
<400> 227
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Thr Asp Tyr
20 25 30
Tyr Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Phe Ile Gly Asn Lys Ala Asn Gly Tyr Thr Thr Glu Tyr Ser Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Thr Arg Asp Arg Gly Leu Arg Phe Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 228
<211> 121
<212> Белок
<213> Искусственная последовательность
<220>
<223> 3-15A5-2
<400> 228
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Thr Asp Tyr
20 25 30
Tyr Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Phe Ile Gly Asn Lys Ala Asn Gly Tyr Thr Thr Glu Tyr Ala Ala
50 55 60
Pro Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Thr Arg Asp Arg Gly Leu Arg Phe Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 229
<211> 121
<212> Белок
<213> Искусственная последовательность
<220>
<223> 3-15A5-3
<400> 229
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Thr Asp Tyr
20 25 30
Tyr Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Phe Ile Gly Asn Lys Ala Asn Gly Gly Thr Thr Asp Tyr Ala Ala
50 55 60
Pro Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Thr Arg Asp Arg Gly Leu Arg Phe Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 230
<211> 95
<212> Белок
<213> Искусственная последовательность
<220>
<223> IGKV3-11
<400> 230
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Arg Ala Thr Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Arg Ser Asn Trp Pro
85 90 95
<210> 231
<211> 106
<212> Белок
<213> Искусственная последовательность
<220>
<223> A5-L1
<400> 231
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Ser Ser Val Thr Tyr Ile
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr
35 40 45
Ala Thr Ser Asn Leu Ala Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu
65 70 75 80
Asp Phe Ala Val Tyr Tyr Cys Gln His Trp Ser Ser Lys Pro Pro Thr
85 90 95
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 232
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> A5-L2
<400> 232
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Tyr
20 25 30
Ile His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Ala Thr Ser Asn Leu Ala Ser Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Gln His Trp Ser Ser Lys Pro Pro
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 233
<211> 106
<212> Белок
<213> Искусственная последовательность
<220>
<223> A5-L3
<400> 233
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Ser Ser Val Thr Tyr Ile
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr
35 40 45
Asp Ala Ser Asn Arg Ala Thr Gly Ile Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu
65 70 75 80
Asp Phe Ala Val Tyr Tyr Cys Gln His Trp Ser Ser Lys Pro Pro Thr
85 90 95
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 234
<211> 106
<212> Белок
<213> Искусственная последовательность
<220>
<223> A5-L4
<400> 234
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Ser Ser Val Thr Tyr Ile
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr
35 40 45
Ala Thr Ser Asn Leu Ala Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu
65 70 75 80
Asp Phe Ala Val Tyr Tyr Cys Gln Gln Trp Ser Ser Lys Pro Pro Thr
85 90 95
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 235
<211> 106
<212> Белок
<213> Искусственная последовательность
<220>
<223> A5-L1A (все обратные мутации)
<400> 235
Gln Thr Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Ser Ser Val Thr Tyr Ile
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Ser Ser Pro Lys Ser Trp Ile Tyr
35 40 45
Ala Thr Ser Asn Leu Ala Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu
65 70 75 80
Asp Phe Ala Val Tyr Tyr Cys Gln His Trp Ser Ser Lys Pro Pro Thr
85 90 95
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 236
<211> 106
<212> Белок
<213> Искусственная последовательность
<220>
<223> A5-L1B (Q1T2)
<400> 236
Gln Thr Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Ser Ser Val Thr Tyr Ile
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr
35 40 45
Ala Thr Ser Asn Leu Ala Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu
65 70 75 80
Asp Phe Ala Val Tyr Tyr Cys Gln His Trp Ser Ser Lys Pro Pro Thr
85 90 95
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 237
<211> 106
<212> Белок
<213> Искусственная последовательность
<220>
<223> A5-L1C (FR2)
<400> 237
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Ser Ser Val Thr Tyr Ile
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Ser Ser Pro Lys Ser Trp Ile Tyr
35 40 45
Ala Thr Ser Asn Leu Ala Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu
65 70 75 80
Asp Phe Ala Val Tyr Tyr Cys Gln His Trp Ser Ser Lys Pro Pro Thr
85 90 95
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 238
<211> 428
<212> Белок
<213> Искусственная последовательность
<220>
<223> NABA-avi-His
<400> 238
Gln Leu Thr Thr Glu Ser Met Pro Phe Asn Val Ala Glu Gly Lys Glu
1 5 10 15
Val Leu Leu Leu Val His Asn Leu Pro Gln Gln Leu Phe Gly Tyr Ser
20 25 30
Trp Tyr Lys Gly Glu Arg Val Asp Gly Asn Arg Gln Ile Val Gly Tyr
35 40 45
Ala Ile Gly Thr Gln Gln Ala Thr Pro Gly Pro Ala Asn Ser Gly Arg
50 55 60
Glu Thr Ile Tyr Pro Asn Ala Ser Leu Leu Ile Gln Asn Val Thr Gln
65 70 75 80
Asn Asp Thr Gly Phe Tyr Thr Leu Gln Val Ile Lys Ser Asp Leu Val
85 90 95
Asn Glu Glu Ala Thr Gly Gln Phe His Val Tyr Pro Glu Leu Pro Lys
100 105 110
Pro Ser Ile Ser Ser Asn Asn Ser Asn Pro Val Glu Asp Lys Asp Ala
115 120 125
Met Ala Phe Thr Cys Glu Pro Glu Thr Gln Asp Thr Thr Tyr Leu Trp
130 135 140
Trp Ile Asn Asn Gln Ser Leu Pro Val Ser Pro Arg Leu Gln Leu Ser
145 150 155 160
Asn Gly Asn Arg Thr Leu Thr Leu Leu Ser Val Thr Arg Asn Asp Thr
165 170 175
Gly Pro Tyr Glu Cys Glu Ile Gln Asn Pro Val Ser Ala Asn Arg Ser
180 185 190
Asp Pro Val Thr Leu Asn Val Thr Tyr Gly Pro Asp Thr Pro Thr Ile
195 200 205
Ser Pro Ser Asp Thr Tyr Tyr Arg Pro Gly Ala Asn Leu Ser Leu Ser
210 215 220
Cys Tyr Ala Ala Ser Asn Pro Pro Ala Gln Tyr Ser Trp Leu Ile Asn
225 230 235 240
Gly Thr Phe Gln Gln Ser Thr Gln Glu Leu Phe Ile Pro Asn Ile Thr
245 250 255
Val Asn Asn Ser Gly Ser Tyr Thr Cys His Ala Asn Asn Ser Val Thr
260 265 270
Gly Cys Asn Arg Thr Thr Val Lys Thr Ile Ile Val Thr Glu Leu Ser
275 280 285
Pro Val Val Ala Lys Pro Gln Ile Lys Ala Ser Lys Thr Thr Val Thr
290 295 300
Gly Asp Lys Asp Ser Val Asn Leu Thr Cys Ser Thr Asn Asp Thr Gly
305 310 315 320
Ile Ser Ile Arg Trp Phe Phe Lys Asn Gln Ser Leu Pro Ser Ser Glu
325 330 335
Arg Met Lys Leu Ser Gln Gly Asn Ile Thr Leu Ser Ile Asn Pro Val
340 345 350
Lys Arg Glu Asp Ala Gly Thr Tyr Trp Cys Glu Val Phe Asn Pro Ile
355 360 365
Ser Lys Asn Gln Ser Asp Pro Ile Met Leu Asn Val Asn Tyr Asn Ala
370 375 380
Leu Pro Gln Glu Asn Leu Ile Asn Val Asp Leu Glu Val Leu Phe Gln
385 390 395 400
Gly Pro Gly Ser Gly Leu Asn Asp Ile Phe Glu Ala Gln Lys Ile Glu
405 410 415
Trp His Glu Ala Arg Ala His His His His His His
420 425
<210> 239
<211> 420
<212> Белок
<213> Искусственная последовательность
<220>
<223> N(A2B2)A-avi-His
<400> 239
Gln Leu Thr Thr Glu Ser Met Pro Phe Asn Val Ala Glu Gly Lys Glu
1 5 10 15
Val Leu Leu Leu Val His Asn Leu Pro Gln Gln Leu Phe Gly Tyr Ser
20 25 30
Trp Tyr Lys Gly Glu Arg Val Asp Gly Asn Arg Gln Ile Val Gly Tyr
35 40 45
Ala Ile Gly Thr Gln Gln Ala Thr Pro Gly Pro Ala Asn Ser Gly Arg
50 55 60
Glu Thr Ile Tyr Pro Asn Ala Ser Leu Leu Ile Gln Asn Val Thr Gln
65 70 75 80
Asn Asp Thr Gly Phe Tyr Thr Leu Gln Val Ile Lys Ser Asp Leu Val
85 90 95
Asn Glu Glu Ala Thr Gly Gln Phe His Val Tyr Pro Glu Leu Pro Lys
100 105 110
Pro Phe Ile Thr Ser Asn Asn Ser Asn Pro Val Glu Asp Glu Asp Ala
115 120 125
Val Ala Leu Thr Cys Glu Pro Glu Ile Gln Asn Thr Thr Tyr Leu Trp
130 135 140
Trp Val Asn Asn Gln Ser Leu Pro Val Ser Pro Arg Leu Gln Leu Ser
145 150 155 160
Asn Asp Asn Arg Thr Leu Thr Leu Leu Ser Val Thr Arg Asn Asp Val
165 170 175
Gly Pro Tyr Glu Cys Gly Ile Gln Asn Lys Leu Ser Val Asp His Ser
180 185 190
Asp Pro Val Ile Leu Asn Val Leu Tyr Gly Pro Asp Asp Pro Thr Ile
195 200 205
Ser Pro Ser Tyr Thr Tyr Tyr Arg Pro Gly Val Asn Leu Ser Leu Ser
210 215 220
Cys His Ala Ala Ser Asn Pro Pro Ala Gln Tyr Ser Trp Leu Ile Asp
225 230 235 240
Gly Asn Ile Gln Gln His Thr Gln Glu Leu Phe Ile Ser Asn Ile Thr
245 250 255
Glu Lys Asn Ser Gly Leu Tyr Thr Cys Gln Ala Asn Asn Ser Ala Ser
260 265 270
Gly His Ser Arg Thr Thr Val Lys Thr Ile Thr Val Ser Ala Leu Ser
275 280 285
Pro Val Val Ala Lys Pro Gln Ile Lys Ala Ser Lys Thr Thr Val Thr
290 295 300
Gly Asp Lys Asp Ser Val Asn Leu Thr Cys Ser Thr Asn Asp Thr Gly
305 310 315 320
Ile Ser Ile Arg Trp Phe Phe Lys Asn Gln Ser Leu Pro Ser Ser Glu
325 330 335
Arg Met Lys Leu Ser Gln Gly Asn Ile Thr Leu Ser Ile Asn Pro Val
340 345 350
Lys Arg Glu Asp Ala Gly Thr Tyr Trp Cys Glu Val Phe Asn Pro Ile
355 360 365
Ser Lys Asn Gln Ser Asp Pro Ile Met Leu Asn Val Asn Tyr Asn Ala
370 375 380
Leu Pro Gln Glu Asn Leu Ile Asn Val Asp Gly Ser Gly Leu Asn Asp
385 390 395 400
Ile Phe Glu Ala Gln Lys Ile Glu Trp His Glu Ala Arg Ala His His
405 410 415
His His His His
420
<210> 240
<211> 420
<212> Белок
<213> Искусственная последовательность
<220>
<223> NA(B2)A-avi-His
<400> 240
Gln Leu Thr Thr Glu Ser Met Pro Phe Asn Val Ala Glu Gly Lys Glu
1 5 10 15
Val Leu Leu Leu Val His Asn Leu Pro Gln Gln Leu Phe Gly Tyr Ser
20 25 30
Trp Tyr Lys Gly Glu Arg Val Asp Gly Asn Arg Gln Ile Val Gly Tyr
35 40 45
Ala Ile Gly Thr Gln Gln Ala Thr Pro Gly Pro Ala Asn Ser Gly Arg
50 55 60
Glu Thr Ile Tyr Pro Asn Ala Ser Leu Leu Ile Gln Asn Val Thr Gln
65 70 75 80
Asn Asp Thr Gly Phe Tyr Thr Leu Gln Val Ile Lys Ser Asp Leu Val
85 90 95
Asn Glu Glu Ala Thr Gly Gln Phe His Val Tyr Pro Glu Leu Pro Lys
100 105 110
Pro Ser Ile Ser Ser Asn Asn Ser Asn Pro Val Glu Asp Lys Asp Ala
115 120 125
Met Ala Phe Thr Cys Glu Pro Glu Thr Gln Asp Thr Thr Tyr Leu Trp
130 135 140
Trp Ile Asn Asn Gln Ser Leu Pro Val Ser Pro Arg Leu Gln Leu Ser
145 150 155 160
Asn Gly Asn Arg Thr Leu Thr Leu Leu Ser Val Thr Arg Asn Asp Thr
165 170 175
Gly Pro Tyr Glu Cys Glu Ile Gln Asn Pro Val Ser Ala Asn Arg Ser
180 185 190
Asp Pro Val Thr Leu Asn Val Thr Tyr Gly Pro Asp Asp Pro Thr Ile
195 200 205
Ser Pro Ser Tyr Thr Tyr Tyr Arg Pro Gly Val Asn Leu Ser Leu Ser
210 215 220
Cys His Ala Ala Ser Asn Pro Pro Ala Gln Tyr Ser Trp Leu Ile Asp
225 230 235 240
Gly Asn Ile Gln Gln His Thr Gln Glu Leu Phe Ile Ser Asn Ile Thr
245 250 255
Glu Lys Asn Ser Gly Leu Tyr Thr Cys Gln Ala Asn Asn Ser Ala Ser
260 265 270
Gly His Ser Arg Thr Thr Val Lys Thr Ile Thr Val Ser Ala Leu Ser
275 280 285
Pro Val Val Ala Lys Pro Gln Ile Lys Ala Ser Lys Thr Thr Val Thr
290 295 300
Gly Asp Lys Asp Ser Val Asn Leu Thr Cys Ser Thr Asn Asp Thr Gly
305 310 315 320
Ile Ser Ile Arg Trp Phe Phe Lys Asn Gln Ser Leu Pro Ser Ser Glu
325 330 335
Arg Met Lys Leu Ser Gln Gly Asn Ile Thr Leu Ser Ile Asn Pro Val
340 345 350
Lys Arg Glu Asp Ala Gly Thr Tyr Trp Cys Glu Val Phe Asn Pro Ile
355 360 365
Ser Lys Asn Gln Ser Asp Pro Ile Met Leu Asn Val Asn Tyr Asn Ala
370 375 380
Leu Pro Gln Glu Asn Leu Ile Asn Val Asp Gly Ser Gly Leu Asn Asp
385 390 395 400
Ile Phe Glu Ala Gln Lys Ile Glu Trp His Glu Ala Arg Ala His His
405 410 415
His His His His
420
<210> 241
<211> 90
<212> ДНК
<213> Искусственная последовательность
<220>
<223> A5H1EL1D_H1_обратн_TN
<220>
<221> примечание
<222> (40)..(42)
<223> n представляет собой a, c, g или t и кодирует 50% N; 20% S; 3%
D/E/Q/G/Y/V/T/H/A/L
<220>
<221> примечание
<222> (46)..(48)
<223> n представляет собой a, c, g или t и кодирует 50% Y; 20% A;
3,75%
G/V/T/H/L/I/R/F
<220>
<221> примечание
<222> (49)..(51)
<223> n представляет собой a, c, g или t и кодирует 60% Y; 4%
G/V/H/S/E/Q/N/D/R/F
<220>
<221> примечание
<222> (52)..(54)
<223> n представляет собой a, c, g или t и кодирует 50% D; 20% S;
4,3%
G/Y/T/N/A/E/Q
<220>
<221> примечание
<222> (55)..(57)
<223> n представляет собой a, c, g или t и кодирует 50% T; 20% S;
4,3%
A/G/Y/N/D/E/Q
<220>
<221> примечание
<222> (61)..(62)
<223> n представляет собой a, c, g или t и кодирует 60% T; 5%
A/S/G/Y/N/D/E/Q
<220>
<221> примечание
<222> (63)..(63)
<223> n представляет собой a, c, g или t
<400> 241
cagccactcg aggcctttac ccggtgcttg gcgtacccan nncatnnnnn nnnnnnngaa
60
nnngaagcca gaagccgcgc agctgagacg
90
<210> 242
<211> 84
<212> ДНК
<213> Искусственная последовательность
<220>
<223> A5H1EL1D_H2_прям_TN
<220>
<221> примечание
<222> (37)..(39)
<223> n представляет собой a, c, g или t и кодирует 60% F; 10% A; 6
% Y/V/L/I/G
<220>
<221> примечание
<222> (43)..(45)
<223> n представляет собой a, c, g или t и кодирует 50% G; 20% S;
3%
A/K/T/V/N/D/E/Q/L/I
<220>
<221> примечание
<222> (46)..(48)
<223> n представляет собой a, c, g или t и кодирует 650% N; 20% G;
3,75 %
D/E/Q/S/Y/T/H/A
<220>
<221> примечание
<222> (49)..(51)
<223> n представляет собой a, c, g или t и кодирует 60% K; 5%
A/T/Y/N/D/E/Q/R
<220>
<221> примечание
<222> (52)..(54)
<223> n представляет собой a, c, g или t и кодирует 60% A; 4%
V/G/D/P/H/N/E/Q/L/I
<220>
<221> примечание
<222> (55)..(57)
<223> n представляет собой a, c, g или t и кодирует 60% N; 5%
D/E/Q; 4,17%
G/T/H/S/A/R
<400> 242
cgccaagcac cgggtaaagg cctcgagtgg ctgggtnnna tcnnnnnnnn nnnnnnngcg
60
tacaccacgg aatactccgc ctcc
84
<210> 243
<211> 26
<212> ДНК
<213> Искусственная последовательность
<220>
<223> LMB3 длинный
<400> 243
caggaaacag ctatgaccat gattac
26
<210> 244
<211> 53
<212> ДНК
<213> Искусственная последовательность
<220>
<223> HCDR3-обратн-констант
<400> 244
aacggtcacc gtggtaccct ggccccagta gtcgaaatag aagcgcagac cac
53
<210> 245
<211> 84
<212> ДНК
<213> Искусственная последовательность
<220>
<223> A5H1EL1D _L1_обратн_TN
<220>
<221> примечание
<222> (37)..(39)
<223> n представляет собой a, c, g или t и кодирует 50% H; 20% A;
3,33%
R/K/G/S/T/Q/Y/N/V
<220>
<221> примечание
<222> (40)..(42)
<223> n представляет собой a, c, g или t и кодирует 70% I; 30% L
<220>
<221> примечание
<222> (43)..(45)
<223> n представляет собой a, c, g или t и кодирует 60% Y; 4%
F/G/A/V/T/H/S/N/Q/R
<220>
<221> примечание
<222> (46)..(48)
<223> n представляет собой a, c, g или t и кодирует 50% T; 20% S;
2,72%
A/G/Y/V/P/H/N/D/E/Q/R
<220>
<221> примечание
<222> (49)..(51)
<223> n представляет собой a, c, g или t и кодирует 50% V; 20% S;
3,33%
T/A/G/N/Q/F/Y/P/H
<220>
<221> примечание
<222> (52)..(54)
<223> n представляет собой a, c, g или t и кодирует 50% S; 20% V;
3,33%
T/A/G/N/D/E/Q/Y/H
<400> 245
ggaacgcggg gcctggcctg gtttttgctg ataccannnn nnnnnnnnnn nnnngctgga
60
tgcgcggcaa gacagggtag cacg
84
<210> 246
<211> 84
<212> ДНК
<213> Искусственная последовательность
<220>
<223> A5H1EL1D _L2_прям_TN
<220>
<221> примечание
<222> (37)..(39)
<223> n представляет собой a, c, g или t и кодирует 60% Y; 10% F;
7,5% H/K/N/S
<220>
<221> примечание
<222> (40)..(42)
<223> n представляет собой a, c, g или t и кодирует 50% A; 20% D;
3,33%
V/G/S/T/Y/H/N/E/Q
<220>
<221> примечание
<222> (43)..(45)
<223> n представляет собой a, c, g или t и кодирует 50% T; 20% A;
3,33%
S/G/V/P/H/N/D/E/Q
<220>
<221> примечание
<222> (46)..(48)
<223> n представляет собой a, c, g или t и кодирует 60% S; 4%
T/A/G/N/D/E/Q/Y/V/H
<220>
<221> примечание
<222> (49)..(51)
<223> n представляет собой a, c, g или t и кодирует 60% N; 4%
D/E/Q/Y/K/T/H/S/A/R
<400> 246
cagcaaaaac caggccaggc cccgcgttcc tggatcnnnn nnnnnnnnnn nctcgcttct
60
ggtatcccgg cacgtttctc cggc
84
<210> 247
<211> 84
<212> ДНК
<213> Искусственная последовательность
<220>
<223> A5H1EL1D _L3_прям_TN
<220>
<221> примечание
<222> (31)..(33)
<223> n представляет собой a, c, g или t и кодирует 90% Q; 10% H
<220>
<221> примечание
<222> (34)..(36)
<223> n представляет собой a, c, g или t и кодирует 60% H; 5%
R/K/Q/E/Y/F/N/D
<220>
<221> примечание
<222> (37)..(39)
<223> n представляет собой a, c, g или t и кодирует 65% W; 7%
F/Y/V/L/I
<220>
<221> примечание
<222> (40)..(42)
<223> n представляет собой a, c, g или t и кодирует 58% S; 4%
T/A/G/N/D/E/Q; 2%
Y/V/P/H/L/I/R
<220>
<221> примечание
<222> (43)..(45)
<223> n представляет собой a, c, g или t и кодирует 58% S; 4%
T/A/G/N/D/E/Q; 2%
Y/V/P/H/L/I/R
<220>
<221> примечание
<222> (46)..(48)
<223> n представляет собой a, c, g или t и кодирует 60% K; 5% R/H;
2,72%
A/V/T/P/Y/N/D/E/Q/L/I
<220>
<221> примечание
<222> (49)..(51)
<223> n представляет собой a, c, g или t и кодирует 70% P; 5%
A/S/T/R/S/L
<220>
<221> примечание
<222> (52)..(54)
<223> n представляет собой a, c, g или t и кодирует 60% P; 5%
L/G/R/M; 2,86%
A/V/L/I/F/S/R
<400> 247
gagcctgaag attttgccgt atactattgt nnnnnnnnnn nnnnnnnnnn nnnnactttc
60
ggtcagggca ccaagctgga aatc
84
<210> 248
<211> 90
<212> ДНК
<213> Искусственная последовательность
<220>
<223> A5H1EL1D _H3_обратн_TN
<220>
<221> примечание
<222> (34)..(36)
<223> n представляет собой a, c, g или t и кодирует 80% F; 10% I/L
<220>
<221> примечание
<222> (37)..(39)
<223> n представляет собой a, c, g или t и кодирует 60% Y; 5% F/W;
2,14%
G/A/V/T/P/H/S/N/D/E/Q/L/I/R
<220>
<221> примечание
<222> (40)..(42)
<223> n представляет собой a, c, g или t и кодирует 65% F; 5%
Y/W/A/V/L/I/G
<220>
<221> примечание
<222> (43)..(45)
<223> n представляет собой a, c, g или t и кодирует 60% R; 5% K/H;
2,72%
A/V/T/P/Y/N/D/E/Q/L/I
<220>
<221> примечание
<222> (46)..(48)
<223> n представляет собой a, c, g или t и кодирует 60% L; 4%
I/V/A/F; 2,4%
G/Y/T/P/H/S/N/D/E/Q
<220>
<221> примечание
<222> (49)..(51)
<223> n представляет собой a, c, g или t и кодирует 60% G; 5% A/S/T;
2,5%
Y/V/P/H/N/D/E/Q/L/I
<220>
<221> примечание
<222> (52)..(54)
<223> n представляет собой a, c, g или t и кодирует 60% R; 5% K/H;
2,72%
A/V/T/P/Y/N/D/E/Q/L/I
<220>
<221> примечание
<222> (55)..(57)
<223> n представляет собой a, c, g или t
<220>
<221> примечание
<222> (58)..(60)
<223> n представляет собой a, c, g или t и кодирует 80% F; 10% I/L
<220>
<221> примечание
<222> (58)..(60)
<223> n представляет собой a, c, g или t и кодирует 60% R; 10% K;
2,72%
A/V/T/P/Y/N/D/E/Q/L/H
<400> 248
aacggtcacc gtggtaccct ggccccagta gtcnnnnnnn nnnnnnnnnn nnnnnnnnnn
60
agtacagtag taggtggcgg tgtcttctgc
90
<210> 249
<211> 30
<212> ДНК
<213> Искусственная последовательность
<220>
<223> LCDR3-обратн-констант
<400> 249
acaatagtat acggcaaaat cttcaggctc
30
<210> 250
<211> 36
<212> ДНК
<213> Искусственная последовательность
<220>
<223> амплификация HCDR3
<400> 250
agaaacggtc accgtggtac cctggcccca gtagtc
36
<210> 251
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P006.038)- CDR-H1
<400> 251
Gly Phe Thr Phe Thr Asp Tyr Tyr Met Asn
1 5 10
<210> 252
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P006.038)- CDR-H2
<400> 252
Phe Ile Gly Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala Ser
1 5 10 15
Val Lys Gly
<210> 253
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P006.038)- CDR-H3
<400> 253
Asp Arg Gly Ile Arg Phe Gly Phe Asp Tyr
1 5 10
<210> 254
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P006.038)- CDR-L1
<400> 254
Arg Ala Ser Ser Ser Val Thr Tyr Ile His
1 5 10
<210> 255
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P006.038)- CDR-L2
<400> 255
Ala Thr Ser Asn Leu Ala Ser
1 5
<210> 256
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P006.038)- CDR-L3
<400> 256
Gln His Trp Ser Ser Val Pro Pro Thr
1 5
<210> 257
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.097)- CDR-H1
<400> 257
Gly Phe Thr Phe Thr Asp Tyr Tyr Met Asn
1 5 10
<210> 258
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.097)- CDR-H2
<400> 258
Phe Ile Gly Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala Ser
1 5 10 15
Val Lys Gly
<210> 259
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.097)- CDR-H3
<400> 259
Asp Arg Gly Leu Arg Phe Ser Phe Asp Tyr
1 5 10
<210> 260
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.097)- CDR-L1
<400> 260
Arg Ala Ser Ser Ser Val Thr Tyr Ile His
1 5 10
<210> 261
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.097)- CDR-L2
<400> 261
Ala Thr Ser Asn Leu Ala Ser
1 5
<210> 262
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.097)- CDR-L3
<400> 262
Gln His Trp Ser Ser Gln Pro Pro Thr
1 5
<210> 263
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.103)- CDR-H1
<400> 263
Gly Phe Thr Phe Thr Asp Tyr Tyr Met Asn
1 5 10
<210> 264
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.103)- CDR-H2
<400> 264
Phe Ile Gly Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala Ser
1 5 10 15
Val Lys Gly
<210> 265
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.103)- CDR-H3
<400> 265
Asp Arg Gly Ile Arg Phe Tyr Phe Asp Tyr
1 5 10
<210> 266
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.103)- CDR-L1
<400> 266
Arg Ala Ser Ser Ser Val Thr Tyr Ile His
1 5 10
<210> 267
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.103)- CDR-L2
<400> 267
Ala Thr Ser Asn Leu Ala Ser
1 5
<210> 268
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.103)- CDR-L3
<400> 268
Gln His Trp Ser Ser Ile Ser Pro Thr
1 5
<210> 269
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P002.139)- CDR-H1
<400> 269
Gly Phe Tyr Phe Thr Asp Tyr Ala Met Asn
1 5 10
<210> 270
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P002.139)- CDR-H2
<400> 270
Val Ile Ser Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala Ser
1 5 10 15
Val Lys Gly
<210> 271
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P002.139)- CDR-H3
<400> 271
Asp Arg Gly Leu Arg Phe Tyr Phe Asp Tyr
1 5 10
<210> 272
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P002.139)- CDR-L1
<400> 272
His Ala Ser Ser Ser Val Thr Tyr Ile His
1 5 10
<210> 273
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P002.139)- CDR-L2
<400> 273
Ala Thr Ser Asn Leu Ala Ser
1 5
<210> 274
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P002.139)- CDR-L3
<400> 274
Gln His Trp Ser Ser Lys Pro Pro Thr
1 5
<210> 275
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P001.177)- CDR-H1
<400> 275
Gly Phe Tyr Phe Thr Asp Tyr Tyr Met Asn
1 5 10
<210> 276
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P001.177)- CDR-H2
<400> 276
Phe Ile Ser Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala Ser
1 5 10 15
Val Lys Gly
<210> 277
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P001.177)- CDR-H3
<400> 277
Asp Arg Gly Leu Arg Phe Tyr Phe Asp Tyr
1 5 10
<210> 278
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P001.177)- CDR-L1
<400> 278
Arg Ala Ser Ser Ser Val Thr Tyr Ile His
1 5 10
<210> 279
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P001.177)- CDR-L2
<400> 279
Ala Thr Ser Asn Leu Ala Ser
1 5
<210> 280
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P001.177)- CDR-L3
<400> 280
Gln His Trp Ser Ser Lys Pro Pro Thr
1 5
<210> 281
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.102)- CDR-H1
<400> 281
Gly Phe Thr Phe Thr Asp Tyr Tyr Met Asn
1 5 10
<210> 282
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.102)- CDR-H2
<400> 282
Phe Ile Gly Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala Ser
1 5 10 15
Val Lys Gly
<210> 283
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.102)- CDR-H3
<400> 283
Asp Arg Gly Ile Arg Phe Gln Phe Asp Tyr
1 5 10
<210> 284
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.102)- CDR-L1
<400> 284
Arg Ala Ser Ser Ser Val Thr Tyr Ile His
1 5 10
<210> 285
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.102)- CDR-L2
<400> 285
Ala Thr Ser Asn Leu Ala Ser
1 5
<210> 286
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.102)- CDR-L3
<400> 286
Gln His Trp Ser Ser Lys Ser Pro Thr
1 5
<210> 287
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.102-комбо1)- CDR-H1
<400> 287
Gly Phe Tyr Phe Thr Asp Tyr Tyr Met Asn
1 5 10
<210> 288
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.102-комбо1)- CDR-H2
<400> 288
Val Ile Ser Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala Ser
1 5 10 15
Val Lys Gly
<210> 289
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.102-комбо1)- CDR-H3
<400> 289
Asp Arg Gly Ile Arg Phe Gln Phe Asp Tyr
1 5 10
<210> 290
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.102-комбо1)- CDR-L1
<400> 290
Arg Ala Ser Ser Ser Val Thr Tyr Ile His
1 5 10
<210> 291
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.102-комбо1)- CDR-L2
<400> 291
Ala Thr Ser Asn Leu Ala Ser
1 5
<210> 292
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.102-комбо1)- CDR-L3
<400> 292
Gln His Trp Ser Ser Lys Ser Pro Thr
1 5
<210> 293
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.102-комбо2)- CDR-H1
<400> 293
Gly Phe Tyr Phe Ser Asp Tyr Tyr Met Asn
1 5 10
<210> 294
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.102-комбо2)- CDR-H2
<400> 294
Val Ile Ser Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala Ser
1 5 10 15
Val Lys Gly
<210> 295
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.102-комбо2)- CDR-H3
<400> 295
Asp Arg Gly Ile Arg Phe Gln Phe Asp Tyr
1 5 10
<210> 296
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.102-комбо2)- CDR-L1
<400> 296
Arg Ala Ser Ser Ser Val Thr Tyr Ile His
1 5 10
<210> 297
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.102-комбо2)- CDR-L2
<400> 297
Ala Thr Ser Asn Leu Ala Ser
1 5
<210> 298
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.102-комбо2)- CDR-L3
<400> 298
Gln His Trp Ser Ser Lys Ser Pro Thr
1 5
<210> 299
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.103-комбо1)- CDR-H1
<400> 299
Gly Phe Thr Phe Thr Asp Tyr Tyr Met Asn
1 5 10
<210> 300
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.103-комбо1)- CDR-H2
<400> 300
Phe Ile Gly Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala Ser
1 5 10 15
Val Lys Gly
<210> 301
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.103-комбо1)- CDR-H3
<400> 301
Asp Arg Gly Ile Arg Phe Ser Phe Asp Tyr
1 5 10
<210> 302
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.103-комбо1)- CDR-L1
<400> 302
Arg Ala Ser Ser Ser Val Thr Tyr Ile His
1 5 10
<210> 303
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.103-комбо1)- CDR-L2
<400> 303
Ala Thr Ser Asn Leu Ala Ser
1 5
<210> 304
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.103-комбо1)- CDR-L3
<400> 304
Gln His Trp Ser Ser Ile Ser Pro Thr
1 5
<210> 305
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.103-комбо2)- CDR-H1
<400> 305
Gly Phe Tyr Phe Thr Asp Tyr Tyr Met Asn
1 5 10
<210> 306
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.103-комбо2)- CDR-H2
<400> 306
Val Ile Ser Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala Ser
1 5 10 15
Val Lys Gly
<210> 307
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.103-комбо2)- CDR-H3
<400> 307
Asp Arg Gly Ile Arg Phe Ser Phe Asp Tyr
1 5 10
<210> 308
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.103-комбо2)- CDR-L1
<400> 308
Arg Ala Ser Ser Ser Val Thr Tyr Ile His
1 5 10
<210> 309
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.103-комбо2)- CDR-L2
<400> 309
Ala Thr Ser Asn Leu Ala Ser
1 5
<210> 310
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P005.103-комбо2)- CDR-L3
<400> 310
Gln His Trp Ser Ser Ile Ser Pro Thr
1 5
<210> 311
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P006.038-комбо1)- CDR-H1
<400> 311
Gly Phe Tyr Phe Thr Asp Tyr Ala Met Asn
1 5 10
<210> 312
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P006.038-комбо1)- CDR-H2
<400> 312
Val Ile Ser Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala Ser
1 5 10 15
Val Lys Gly
<210> 313
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P006.038-комбо1)- CDR-H3
<400> 313
Asp Arg Gly Ile Arg Phe Gly Phe Asp Tyr
1 5 10
<210> 314
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P006.038-комбо1)- CDR-L1
<400> 314
Arg Ala Ser Ser Ser Val Thr Tyr Ile His
1 5 10
<210> 315
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P006.038-комбо1)- CDR-L2
<400> 315
Ala Thr Ser Asn Leu Ala Ser
1 5
<210> 316
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P006.038-комбо1)- CDR-L3
<400> 316
Gln His Trp Ser Ser Val Pro Pro Thr
1 5
<210> 317
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P006.038-комбо2)- CDR-H1
<400> 317
Gly Phe Thr Phe Ser Asp Tyr Glu Met Asn
1 5 10
<210> 318
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P006.038-комбо2)- CDR-H2
<400> 318
Phe Ile Ser Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala Ser
1 5 10 15
Val Lys Gly
<210> 319
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P006.038-комбо2)- CDR-H3
<400> 319
Asp Arg Gly Ile Arg Phe Gly Phe Asp Tyr
1 5 10
<210> 320
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P006.038-комбо2)- CDR-L1
<400> 320
Arg Ala Ser Ser Ser Val Thr Tyr Ile His
1 5 10
<210> 321
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P006.038-комбо2)- CDR-L2
<400> 321
Ala Thr Ser Asn Leu Ala Ser
1 5
<210> 322
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (P006.038-комбо2)- CDR-L3
<400> 322
Gln His Trp Ser Ser Val Pro Pro Thr
1 5
<210> 323
<211> 449
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL CEA (A5H1EL1D) -CH1- Fc-впадина PGLALA
<400> 323
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Thr Asp Tyr
20 25 30
Tyr Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Phe Ile Gly Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Lys Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr
85 90 95
Tyr Cys Thr Arg Asp Arg Gly Leu Arg Phe Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
210 215 220
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly
225 230 235 240
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
245 250 255
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
260 265 270
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
275 280 285
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
290 295 300
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
305 310 315 320
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile
325 330 335
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
340 345 350
Cys Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser
355 360 365
Leu Ser Cys Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
370 375 380
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
385 390 395 400
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val
405 410 415
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
420 425 430
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
435 440 445
Pro
<210> 324
<211> 213
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH CEA (A5H1EL1D) - CL
<400> 324
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Ser Ser Val Thr Tyr Ile
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Ser Trp Ile Tyr
35 40 45
Ala Thr Ser Asn Leu Ala Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu
65 70 75 80
Asp Phe Ala Val Tyr Tyr Cys Gln His Trp Ser Ser Lys Pro Pro Thr
85 90 95
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Arg Thr Val Ala Ala Pro
100 105 110
Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr
115 120 125
Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys
130 135 140
Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu
145 150 155 160
Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser
165 170 175
Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala
180 185 190
Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe
195 200 205
Asn Arg Gly Glu Cys
210
<210> 325
<211> 436
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL CEA (P006.038) -CH1- Fc-впадина PGLALA
<400> 325
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Ser Ser Val Thr Tyr Ile
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Ser Trp Ile Tyr
35 40 45
Ala Thr Ser Asn Leu Ala Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu
65 70 75 80
Asp Phe Ala Val Tyr Tyr Cys Gln His Trp Ser Ser Val Pro Pro Thr
85 90 95
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Ser Ser Ala Ser Thr Lys
100 105 110
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
115 120 125
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
130 135 140
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
145 150 155 160
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
165 170 175
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
180 185 190
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro
195 200 205
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
210 215 220
Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
225 230 235 240
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
245 250 255
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
260 265 270
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
275 280 285
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
290 295 300
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly
305 310 315 320
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
325 330 335
Pro Gln Val Cys Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn
340 345 350
Gln Val Ser Leu Ser Cys Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile
355 360 365
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
370 375 380
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Val Ser Lys
385 390 395 400
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
405 410 415
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
420 425 430
Ser Leu Ser Pro
435
<210> 326
<211> 228
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH CEA (P006.038) - CL
<400> 326
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Thr Asp Tyr
20 25 30
Tyr Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Phe Ile Gly Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Lys Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr
85 90 95
Tyr Cys Thr Arg Asp Arg Gly Ile Arg Phe Gly Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Val Ala Ala Pro Ser
115 120 125
Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala
130 135 140
Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val
145 150 155 160
Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser
165 170 175
Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr
180 185 190
Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys
195 200 205
Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn
210 215 220
Arg Gly Glu Cys
225
<210> 327
<211> 436
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL CEA (P005.097) -CH1- Fc-впадина PGLALA
<400> 327
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Ser Ser Val Thr Tyr Ile
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Ser Trp Ile Tyr
35 40 45
Ala Thr Ser Asn Leu Ala Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu
65 70 75 80
Asp Phe Ala Val Tyr Tyr Cys Gln His Trp Ser Ser Gln Pro Pro Thr
85 90 95
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Ser Ser Ala Ser Thr Lys
100 105 110
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
115 120 125
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
130 135 140
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
145 150 155 160
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
165 170 175
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
180 185 190
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro
195 200 205
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
210 215 220
Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
225 230 235 240
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
245 250 255
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
260 265 270
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
275 280 285
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
290 295 300
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly
305 310 315 320
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
325 330 335
Pro Gln Val Cys Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn
340 345 350
Gln Val Ser Leu Ser Cys Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile
355 360 365
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
370 375 380
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Val Ser Lys
385 390 395 400
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
405 410 415
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
420 425 430
Ser Leu Ser Pro
435
<210> 328
<211> 228
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH CEA (P005.097) - CL
<400> 328
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Thr Asp Tyr
20 25 30
Tyr Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Phe Ile Gly Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Lys Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr
85 90 95
Tyr Cys Thr Arg Asp Arg Gly Leu Arg Phe Ser Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Val Ala Ala Pro Ser
115 120 125
Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala
130 135 140
Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val
145 150 155 160
Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser
165 170 175
Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr
180 185 190
Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys
195 200 205
Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn
210 215 220
Arg Gly Glu Cys
225
<210> 329
<211> 436
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL CEA (P005.103) -CH1- Fc-впадина PGLALA
<400> 329
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Ser Ser Val Thr Tyr Ile
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Ser Trp Ile Tyr
35 40 45
Ala Thr Ser Asn Leu Ala Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu
65 70 75 80
Asp Phe Ala Val Tyr Tyr Cys Gln His Trp Ser Ser Ile Ser Pro Thr
85 90 95
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Ser Ser Ala Ser Thr Lys
100 105 110
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
115 120 125
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
130 135 140
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
145 150 155 160
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
165 170 175
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
180 185 190
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro
195 200 205
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
210 215 220
Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
225 230 235 240
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
245 250 255
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
260 265 270
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
275 280 285
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
290 295 300
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly
305 310 315 320
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
325 330 335
Pro Gln Val Cys Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn
340 345 350
Gln Val Ser Leu Ser Cys Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile
355 360 365
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
370 375 380
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Val Ser Lys
385 390 395 400
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
405 410 415
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
420 425 430
Ser Leu Ser Pro
435
<210> 330
<211> 228
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH CEA (P005.103) - CL
<400> 330
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Thr Asp Tyr
20 25 30
Tyr Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Phe Ile Gly Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Lys Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr
85 90 95
Tyr Cys Thr Arg Asp Arg Gly Ile Arg Phe Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Val Ala Ala Pro Ser
115 120 125
Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala
130 135 140
Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val
145 150 155 160
Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser
165 170 175
Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr
180 185 190
Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys
195 200 205
Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn
210 215 220
Arg Gly Glu Cys
225
<210> 331
<211> 436
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL CEA (P002.139) -CH1- Fc-впадина PGLALA
<400> 331
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys His Ala Ser Ser Ser Val Thr Tyr Ile
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Ser Trp Ile Tyr
35 40 45
Ala Thr Ser Asn Leu Ala Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu
65 70 75 80
Asp Phe Ala Val Tyr Tyr Cys Gln His Trp Ser Ser Lys Pro Pro Thr
85 90 95
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Ser Ser Ala Ser Thr Lys
100 105 110
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
115 120 125
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
130 135 140
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
145 150 155 160
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
165 170 175
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
180 185 190
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro
195 200 205
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
210 215 220
Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
225 230 235 240
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
245 250 255
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
260 265 270
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
275 280 285
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
290 295 300
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly
305 310 315 320
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
325 330 335
Pro Gln Val Cys Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn
340 345 350
Gln Val Ser Leu Ser Cys Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile
355 360 365
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
370 375 380
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Val Ser Lys
385 390 395 400
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
405 410 415
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
420 425 430
Ser Leu Ser Pro
435
<210> 332
<211> 228
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH CEA (P002.139) - CL
<400> 332
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Tyr Phe Thr Asp Tyr
20 25 30
Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Val Ile Ser Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Lys Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr
85 90 95
Tyr Cys Thr Arg Asp Arg Gly Leu Arg Phe Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Val Ala Ala Pro Ser
115 120 125
Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala
130 135 140
Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val
145 150 155 160
Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser
165 170 175
Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr
180 185 190
Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys
195 200 205
Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn
210 215 220
Arg Gly Glu Cys
225
<210> 333
<211> 436
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL CEA (P001.177) -CH1- Fc-впадина PGLALA
<400> 333
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Ser Ser Val Thr Tyr Ile
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Ser Trp Ile Tyr
35 40 45
Ala Thr Ser Asn Leu Ala Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu
65 70 75 80
Asp Phe Ala Val Tyr Tyr Cys Gln His Trp Ser Ser Lys Pro Pro Thr
85 90 95
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Ser Ser Ala Ser Thr Lys
100 105 110
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
115 120 125
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
130 135 140
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
145 150 155 160
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
165 170 175
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
180 185 190
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro
195 200 205
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
210 215 220
Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
225 230 235 240
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
245 250 255
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
260 265 270
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
275 280 285
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
290 295 300
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly
305 310 315 320
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
325 330 335
Pro Gln Val Cys Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn
340 345 350
Gln Val Ser Leu Ser Cys Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile
355 360 365
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
370 375 380
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Val Ser Lys
385 390 395 400
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
405 410 415
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
420 425 430
Ser Leu Ser Pro
435
<210> 334
<211> 228
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH CEA (P001.177) - CL
<400> 334
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Tyr Phe Thr Asp Tyr
20 25 30
Tyr Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Phe Ile Ser Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Lys Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr
85 90 95
Tyr Cys Thr Arg Asp Arg Gly Leu Arg Phe Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Val Ala Ala Pro Ser
115 120 125
Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala
130 135 140
Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val
145 150 155 160
Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser
165 170 175
Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr
180 185 190
Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys
195 200 205
Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn
210 215 220
Arg Gly Glu Cys
225
<210> 335
<211> 436
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL CEA (P005.102) -CH1- Fc-впадина PGLALA
<400> 335
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Ser Ser Val Thr Tyr Ile
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Ser Trp Ile Tyr
35 40 45
Ala Thr Ser Asn Leu Ala Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu
65 70 75 80
Asp Phe Ala Val Tyr Tyr Cys Gln His Trp Ser Ser Lys Ser Pro Thr
85 90 95
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Ser Ser Ala Ser Thr Lys
100 105 110
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
115 120 125
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
130 135 140
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
145 150 155 160
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
165 170 175
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
180 185 190
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro
195 200 205
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
210 215 220
Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
225 230 235 240
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
245 250 255
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
260 265 270
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
275 280 285
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
290 295 300
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly
305 310 315 320
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
325 330 335
Pro Gln Val Cys Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn
340 345 350
Gln Val Ser Leu Ser Cys Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile
355 360 365
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
370 375 380
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Val Ser Lys
385 390 395 400
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
405 410 415
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
420 425 430
Ser Leu Ser Pro
435
<210> 336
<211> 228
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH CEA (P005.102) - CL
<400> 336
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Thr Asp Tyr
20 25 30
Tyr Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Phe Ile Gly Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Lys Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr
85 90 95
Tyr Cys Thr Arg Asp Arg Gly Ile Arg Phe Gln Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Val Ala Ala Pro Ser
115 120 125
Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala
130 135 140
Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val
145 150 155 160
Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser
165 170 175
Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr
180 185 190
Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys
195 200 205
Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn
210 215 220
Arg Gly Glu Cys
225
<210> 337
<211> 436
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL CEA (P005.102 комбо1) -CH1- Fc-впадина PGLALA
<400> 337
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Ser Ser Val Thr Tyr Ile
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Ser Trp Ile Tyr
35 40 45
Ala Thr Ser Asn Leu Ala Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu
65 70 75 80
Asp Phe Ala Val Tyr Tyr Cys Gln His Trp Ser Ser Lys Ser Pro Thr
85 90 95
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Ser Ser Ala Ser Thr Lys
100 105 110
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
115 120 125
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
130 135 140
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
145 150 155 160
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
165 170 175
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
180 185 190
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro
195 200 205
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
210 215 220
Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
225 230 235 240
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
245 250 255
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
260 265 270
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
275 280 285
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
290 295 300
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly
305 310 315 320
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
325 330 335
Pro Gln Val Cys Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn
340 345 350
Gln Val Ser Leu Ser Cys Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile
355 360 365
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
370 375 380
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Val Ser Lys
385 390 395 400
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
405 410 415
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
420 425 430
Ser Leu Ser Pro
435
<210> 338
<211> 228
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH CEA (P005.102 комбо1) - CL
<400> 338
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Tyr Phe Thr Asp Tyr
20 25 30
Tyr Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Val Ile Ser Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Lys Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr
85 90 95
Tyr Cys Thr Arg Asp Arg Gly Ile Arg Phe Gln Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Val Ala Ala Pro Ser
115 120 125
Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala
130 135 140
Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val
145 150 155 160
Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser
165 170 175
Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr
180 185 190
Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys
195 200 205
Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn
210 215 220
Arg Gly Glu Cys
225
<210> 339
<211> 436
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL CEA (P005.102 комбо2) -CH1- Fc-впадина PGLALA
<400> 339
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Ser Ser Val Thr Tyr Ile
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Ser Trp Ile Tyr
35 40 45
Ala Thr Ser Asn Leu Ala Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu
65 70 75 80
Asp Phe Ala Val Tyr Tyr Cys Gln His Trp Ser Ser Lys Ser Pro Thr
85 90 95
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Ser Ser Ala Ser Thr Lys
100 105 110
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
115 120 125
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
130 135 140
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
145 150 155 160
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
165 170 175
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
180 185 190
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro
195 200 205
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
210 215 220
Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
225 230 235 240
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
245 250 255
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
260 265 270
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
275 280 285
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
290 295 300
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly
305 310 315 320
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
325 330 335
Pro Gln Val Cys Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn
340 345 350
Gln Val Ser Leu Ser Cys Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile
355 360 365
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
370 375 380
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Val Ser Lys
385 390 395 400
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
405 410 415
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
420 425 430
Ser Leu Ser Pro
435
<210> 340
<211> 228
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH CEA (P005.102 комбо2) - CL
<400> 340
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Tyr Phe Ser Asp Tyr
20 25 30
Tyr Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Val Ile Ser Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Lys Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr
85 90 95
Tyr Cys Thr Arg Asp Arg Gly Ile Arg Phe Gln Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Val Ala Ala Pro Ser
115 120 125
Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala
130 135 140
Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val
145 150 155 160
Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser
165 170 175
Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr
180 185 190
Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys
195 200 205
Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn
210 215 220
Arg Gly Glu Cys
225
<210> 341
<211> 436
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL CEA (P005.103 комбо1) -CH1- Fc-впадина PGLALA
<400> 341
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Ser Ser Val Thr Tyr Ile
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Ser Trp Ile Tyr
35 40 45
Ala Thr Ser Asn Leu Ala Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu
65 70 75 80
Asp Phe Ala Val Tyr Tyr Cys Gln His Trp Ser Ser Ile Ser Pro Thr
85 90 95
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Ser Ser Ala Ser Thr Lys
100 105 110
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
115 120 125
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
130 135 140
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
145 150 155 160
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
165 170 175
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
180 185 190
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro
195 200 205
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
210 215 220
Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
225 230 235 240
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
245 250 255
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
260 265 270
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
275 280 285
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
290 295 300
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly
305 310 315 320
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
325 330 335
Pro Gln Val Cys Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn
340 345 350
Gln Val Ser Leu Ser Cys Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile
355 360 365
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
370 375 380
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Val Ser Lys
385 390 395 400
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
405 410 415
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
420 425 430
Ser Leu Ser Pro
435
<210> 342
<211> 228
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH CEA (P005.103 комбо1) - CL
<400> 342
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Thr Asp Tyr
20 25 30
Tyr Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Phe Ile Gly Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Lys Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr
85 90 95
Tyr Cys Thr Arg Asp Arg Gly Ile Arg Phe Ser Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Val Ala Ala Pro Ser
115 120 125
Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala
130 135 140
Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val
145 150 155 160
Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser
165 170 175
Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr
180 185 190
Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys
195 200 205
Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn
210 215 220
Arg Gly Glu Cys
225
<210> 343
<211> 436
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL CEA (P005.103 комбо2) -CH1- Fc-впадина PGLALA
<400> 343
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Ser Ser Val Thr Tyr Ile
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Ser Trp Ile Tyr
35 40 45
Ala Thr Ser Asn Leu Ala Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu
65 70 75 80
Asp Phe Ala Val Tyr Tyr Cys Gln His Trp Ser Ser Ile Ser Pro Thr
85 90 95
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Ser Ser Ala Ser Thr Lys
100 105 110
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
115 120 125
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
130 135 140
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
145 150 155 160
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
165 170 175
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
180 185 190
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro
195 200 205
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
210 215 220
Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
225 230 235 240
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
245 250 255
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
260 265 270
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
275 280 285
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
290 295 300
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly
305 310 315 320
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
325 330 335
Pro Gln Val Cys Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn
340 345 350
Gln Val Ser Leu Ser Cys Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile
355 360 365
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
370 375 380
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Val Ser Lys
385 390 395 400
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
405 410 415
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
420 425 430
Ser Leu Ser Pro
435
<210> 344
<211> 228
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH CEA (P005.103 комбо2) - CL
<400> 344
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Tyr Phe Thr Asp Tyr
20 25 30
Tyr Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Val Ile Ser Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Lys Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr
85 90 95
Tyr Cys Thr Arg Asp Arg Gly Ile Arg Phe Ser Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Val Ala Ala Pro Ser
115 120 125
Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala
130 135 140
Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val
145 150 155 160
Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser
165 170 175
Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr
180 185 190
Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys
195 200 205
Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn
210 215 220
Arg Gly Glu Cys
225
<210> 345
<211> 436
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL CEA (P006.038 комбо1) -CH1- Fc-впадина PGLALA
<400> 345
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Ser Ser Val Thr Tyr Ile
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Ser Trp Ile Tyr
35 40 45
Ala Thr Ser Asn Leu Ala Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu
65 70 75 80
Asp Phe Ala Val Tyr Tyr Cys Gln His Trp Ser Ser Val Pro Pro Thr
85 90 95
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Ser Ser Ala Ser Thr Lys
100 105 110
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
115 120 125
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
130 135 140
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
145 150 155 160
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
165 170 175
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
180 185 190
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro
195 200 205
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
210 215 220
Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
225 230 235 240
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
245 250 255
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
260 265 270
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
275 280 285
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
290 295 300
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly
305 310 315 320
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
325 330 335
Pro Gln Val Cys Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn
340 345 350
Gln Val Ser Leu Ser Cys Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile
355 360 365
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
370 375 380
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Val Ser Lys
385 390 395 400
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
405 410 415
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
420 425 430
Ser Leu Ser Pro
435
<210> 346
<211> 228
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH CEA (P006.038 комбо1) - CL
<400> 346
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Tyr Phe Thr Asp Tyr
20 25 30
Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Val Ile Ser Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Lys Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr
85 90 95
Tyr Cys Thr Arg Asp Arg Gly Ile Arg Phe Gly Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Val Ala Ala Pro Ser
115 120 125
Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala
130 135 140
Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val
145 150 155 160
Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser
165 170 175
Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr
180 185 190
Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys
195 200 205
Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn
210 215 220
Arg Gly Glu Cys
225
<210> 347
<211> 436
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL CEA (P006.038 комбо2) -CH1- Fc-впадина PGLALA
<400> 347
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Ser Ser Val Thr Tyr Ile
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Ser Trp Ile Tyr
35 40 45
Ala Thr Ser Asn Leu Ala Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu
65 70 75 80
Asp Phe Ala Val Tyr Tyr Cys Gln His Trp Ser Ser Val Pro Pro Thr
85 90 95
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Ser Ser Ala Ser Thr Lys
100 105 110
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
115 120 125
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
130 135 140
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
145 150 155 160
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
165 170 175
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
180 185 190
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro
195 200 205
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
210 215 220
Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
225 230 235 240
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
245 250 255
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
260 265 270
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
275 280 285
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
290 295 300
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly
305 310 315 320
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
325 330 335
Pro Gln Val Cys Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn
340 345 350
Gln Val Ser Leu Ser Cys Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile
355 360 365
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
370 375 380
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Val Ser Lys
385 390 395 400
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
405 410 415
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
420 425 430
Ser Leu Ser Pro
435
<210> 348
<211> 228
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH CEA (P006.038 комбо2) - CL
<400> 348
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Tyr
20 25 30
Glu Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Phe Ile Ser Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Lys Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr
85 90 95
Tyr Cys Thr Arg Asp Arg Gly Ile Arg Phe Gly Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Val Ala Ala Pro Ser
115 120 125
Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala
130 135 140
Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val
145 150 155 160
Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser
165 170 175
Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr
180 185 190
Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys
195 200 205
Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn
210 215 220
Arg Gly Glu Cys
225
<210> 349
<211> 448
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH CD28 (SA_Variant 15) - CH1- Fc-выступ PGLALA
<400> 349
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Ser Ile Tyr Pro Gly Asn Val Gln Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Trp Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Glu Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Glu Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
<210> 350
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL CD28 (SA_Variant 15) - CL
<400> 350
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys His Ala Ser Gln Asn Ile Tyr Val Phe
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gly Gln Thr Tyr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Arg Lys Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 351
<211> 436
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA(A5H1EL1D) VL-CH1 hu IgG1 Fc-впадина PGLALA
<400> 351
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Ser Ser Val Thr Tyr Ile
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Ser Trp Ile Tyr
35 40 45
Ala Thr Ser Asn Leu Ala Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu
65 70 75 80
Asp Phe Ala Val Tyr Tyr Cys Gln His Trp Ser Ser Lys Pro Pro Thr
85 90 95
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Ser Ser Ala Ser Thr Lys
100 105 110
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
115 120 125
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
130 135 140
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
145 150 155 160
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
165 170 175
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
180 185 190
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro
195 200 205
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
210 215 220
Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
225 230 235 240
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
245 250 255
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
260 265 270
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
275 280 285
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
290 295 300
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly
305 310 315 320
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
325 330 335
Pro Gln Val Cys Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn
340 345 350
Gln Val Ser Leu Ser Cys Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile
355 360 365
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
370 375 380
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Val Ser Lys
385 390 395 400
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
405 410 415
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
420 425 430
Ser Leu Ser Pro
435
<210> 352
<211> 228
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA(A5H1EL1D) VH-C-каппа
<400> 352
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Thr Asp Tyr
20 25 30
Tyr Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Phe Ile Gly Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Lys Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr
85 90 95
Tyr Cys Thr Arg Asp Arg Gly Leu Arg Phe Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Val Ala Ala Pro Ser
115 120 125
Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala
130 135 140
Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val
145 150 155 160
Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser
165 170 175
Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr
180 185 190
Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys
195 200 205
Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn
210 215 220
Arg Gly Glu Cys
225
<210> 353
<211> 448
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA) hu IgG1 VH-CH1 "EE" Fc-выступ PGLALA
<400> 353
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Cys Ile Tyr Pro Gly Asn Val Asn Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Trp Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Glu Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Glu Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
<210> 354
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA) hu IgG1 VL-Ck "RK"
<400> 354
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys His Ala Ser Gln Asn Ile Tyr Val Trp
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gly Gln Thr Tyr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Arg Lys Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 355
<211> 448
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA_Variant 8) hu IgG1 VH-CH1 "EE" Fc-выступ PGLALA
<400> 355
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Ser Ile Tyr Pro Gly Asn Val Gln Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Phe Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Glu Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Glu Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
<210> 356
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA_Variant 8) hu IgG1 VL-Ck "RK"
<400> 356
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys His Ala Ser Gln Asn Ile Tyr Val Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gly Gln Thr Tyr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Arg Lys Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 357
<211> 448
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA_Variant 15) hu IgG1 VH-CH1 "EE" Fc-выступ PGLALA
<400> 357
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Ser Ile Tyr Pro Gly Asn Val Gln Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Trp Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Glu Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Glu Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
<210> 358
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA_Variant 15) hu IgG1 VL-Ck "RK"
<400> 358
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys His Ala Ser Gln Asn Ile Tyr Val Phe
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gly Gln Thr Tyr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Arg Lys Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 359
<211> 448
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA_Variant 29) hu IgG1 VH-CH1 "EE" Fc-выступ PGLALA
<400> 359
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Ser Ile Tyr Pro Gly Asn Val Asn Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Trp Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Glu Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Glu Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
<210> 360
<211> 449
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA(A5H1EL1D) hu IgG1 VH-CH1 "EE" Fc-впадина PGLALA
<400> 360
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Thr Asp Tyr
20 25 30
Tyr Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Phe Ile Gly Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Lys Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr
85 90 95
Tyr Cys Thr Arg Asp Arg Gly Leu Arg Phe Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Glu Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Glu Lys Val Glu Pro Lys Ser Cys
210 215 220
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly
225 230 235 240
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
245 250 255
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
260 265 270
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
275 280 285
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
290 295 300
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
305 310 315 320
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile
325 330 335
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
340 345 350
Cys Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser
355 360 365
Leu Ser Cys Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
370 375 380
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
385 390 395 400
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val
405 410 415
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
420 425 430
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
435 440 445
Pro
<210> 361
<211> 213
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA(A5H1EL1D) hu IgG1 VL-Ck "RK"
<400> 361
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Ser Ser Val Thr Tyr Ile
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Ser Trp Ile Tyr
35 40 45
Ala Thr Ser Asn Leu Ala Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu
65 70 75 80
Asp Phe Ala Val Tyr Tyr Cys Gln His Trp Ser Ser Lys Pro Pro Thr
85 90 95
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Arg Thr Val Ala Ala Pro
100 105 110
Ser Val Phe Ile Phe Pro Pro Ser Asp Arg Lys Leu Lys Ser Gly Thr
115 120 125
Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys
130 135 140
Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu
145 150 155 160
Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser
165 170 175
Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala
180 185 190
Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe
195 200 205
Asn Arg Gly Glu Cys
210
<210> 362
<211> 437
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA) VL-CH1 hu IgG1 Fc-выступ PGLALA
<400> 362
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys His Ala Ser Gln Asn Ile Tyr Val Trp
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gly Gln Thr Tyr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Ser Ser Ala Ser Thr
100 105 110
Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser
115 120 125
Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu
130 135 140
Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His
145 150 155 160
Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser
165 170 175
Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys
180 185 190
Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu
195 200 205
Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro
210 215 220
Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
225 230 235 240
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
245 250 255
Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp
260 265 270
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr
275 280 285
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
290 295 300
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu
305 310 315 320
Gly Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
325 330 335
Glu Pro Gln Val Tyr Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys
340 345 350
Asn Gln Val Ser Leu Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
355 360 365
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
370 375 380
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
385 390 395 400
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser
405 410 415
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
420 425 430
Leu Ser Leu Ser Pro
435
<210> 363
<211> 227
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA) VH-C-каппа
<400> 363
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Cys Ile Tyr Pro Gly Asn Val Asn Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Trp Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Val Ala Ala Pro Ser Val
115 120 125
Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser
130 135 140
Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln
145 150 155 160
Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val
165 170 175
Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu
180 185 190
Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu
195 200 205
Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg
210 215 220
Gly Glu Cys
225
<210> 364
<211> 437
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA_Variant 8) VL-CH1 hu IgG1 Fc-выступ PGLALA
<400> 364
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys His Ala Ser Gln Asn Ile Tyr Val Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gly Gln Thr Tyr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Ser Ser Ala Ser Thr
100 105 110
Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser
115 120 125
Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu
130 135 140
Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His
145 150 155 160
Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser
165 170 175
Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys
180 185 190
Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu
195 200 205
Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro
210 215 220
Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
225 230 235 240
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
245 250 255
Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp
260 265 270
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr
275 280 285
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
290 295 300
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu
305 310 315 320
Gly Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
325 330 335
Glu Pro Gln Val Tyr Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys
340 345 350
Asn Gln Val Ser Leu Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
355 360 365
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
370 375 380
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
385 390 395 400
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser
405 410 415
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
420 425 430
Leu Ser Leu Ser Pro
435
<210> 365
<211> 227
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA_Variant 8) VH-C-каппа
<400> 365
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Ser Ile Tyr Pro Gly Asn Val Gln Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Phe Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Val Ala Ala Pro Ser Val
115 120 125
Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser
130 135 140
Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln
145 150 155 160
Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val
165 170 175
Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu
180 185 190
Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu
195 200 205
Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg
210 215 220
Gly Glu Cys
225
<210> 366
<211> 437
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA_Variant 15) VL-CH1 hu IgG1 Fc-выступ PGLALA
<400> 366
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys His Ala Ser Gln Asn Ile Tyr Val Phe
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gly Gln Thr Tyr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Ser Ser Ala Ser Thr
100 105 110
Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser
115 120 125
Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu
130 135 140
Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His
145 150 155 160
Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser
165 170 175
Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys
180 185 190
Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu
195 200 205
Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro
210 215 220
Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
225 230 235 240
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
245 250 255
Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp
260 265 270
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr
275 280 285
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
290 295 300
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu
305 310 315 320
Gly Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
325 330 335
Glu Pro Gln Val Tyr Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys
340 345 350
Asn Gln Val Ser Leu Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
355 360 365
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
370 375 380
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
385 390 395 400
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser
405 410 415
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
420 425 430
Leu Ser Leu Ser Pro
435
<210> 367
<211> 227
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA_Variant 15) VH-C-каппа
<400> 367
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Ser Ile Tyr Pro Gly Asn Val Gln Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Trp Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Val Ala Ala Pro Ser Val
115 120 125
Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser
130 135 140
Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln
145 150 155 160
Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val
165 170 175
Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu
180 185 190
Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu
195 200 205
Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg
210 215 220
Gly Glu Cys
225
<210> 368
<211> 227
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA_Variant 29) VH-C-каппа
<400> 368
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Ser Ile Tyr Pro Gly Asn Val Asn Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Trp Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Val Ala Ala Pro Ser Val
115 120 125
Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser
130 135 140
Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln
145 150 155 160
Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val
165 170 175
Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu
180 185 190
Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu
195 200 205
Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg
210 215 220
Gly Glu Cys
225
<210> 369
<211> 441
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA(T84.66) VL-CH1 hu IgG1 Fc-впадина PGLALA
<400> 369
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Gly Glu Ser Val Asp Ile Phe
20 25 30
Gly Val Gly Phe Leu His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro
35 40 45
Arg Leu Leu Ile Tyr Arg Ala Ser Asn Arg Ala Thr Gly Ile Pro Ala
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
65 70 75 80
Ser Leu Glu Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Thr Asn
85 90 95
Glu Asp Pro Tyr Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Ser
100 105 110
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser
115 120 125
Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
130 135 140
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
145 150 155 160
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
165 170 175
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln
180 185 190
Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp
195 200 205
Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro
210 215 220
Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro
225 230 235 240
Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr
245 250 255
Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn
260 265 270
Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg
275 280 285
Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val
290 295 300
Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser
305 310 315 320
Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys
325 330 335
Gly Gln Pro Arg Glu Pro Gln Val Cys Thr Leu Pro Pro Ser Arg Asp
340 345 350
Glu Leu Thr Lys Asn Gln Val Ser Leu Ser Cys Ala Val Lys Gly Phe
355 360 365
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
370 375 380
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
385 390 395 400
Phe Leu Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly
405 410 415
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
420 425 430
Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440
<210> 370
<211> 228
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA(T84.66) VH-C-каппа
<400> 370
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Phe Asn Ile Lys Asp Thr
20 25 30
Tyr Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Arg Ile Asp Pro Ala Asn Gly Asn Ser Lys Tyr Val Pro Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Pro Phe Gly Tyr Tyr Val Ser Asp Tyr Ala Met Ala Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Val Ala Ala Pro Ser
115 120 125
Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala
130 135 140
Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val
145 150 155 160
Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser
165 170 175
Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr
180 185 190
Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys
195 200 205
Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn
210 215 220
Arg Gly Glu Cys
225
<210> 371
<211> 449
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA(T84.66) hu IgG1 VH-CH1 "EE" Fc-впадина PGLALA
<400> 371
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Phe Asn Ile Lys Asp Thr
20 25 30
Tyr Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Arg Ile Asp Pro Ala Asn Gly Asn Ser Lys Tyr Val Pro Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Pro Phe Gly Tyr Tyr Val Ser Asp Tyr Ala Met Ala Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Glu Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Glu Lys Val Glu Pro Lys Ser Cys
210 215 220
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly
225 230 235 240
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
245 250 255
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
260 265 270
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
275 280 285
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
290 295 300
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
305 310 315 320
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile
325 330 335
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
340 345 350
Cys Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser
355 360 365
Leu Ser Cys Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
370 375 380
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
385 390 395 400
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val
405 410 415
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
420 425 430
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
435 440 445
Pro
<210> 372
<211> 218
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA(T84.66) hu IgG1 VL-Ck "RK"
<400> 372
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Gly Glu Ser Val Asp Ile Phe
20 25 30
Gly Val Gly Phe Leu His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro
35 40 45
Arg Leu Leu Ile Tyr Arg Ala Ser Asn Arg Ala Thr Gly Ile Pro Ala
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
65 70 75 80
Ser Leu Glu Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Thr Asn
85 90 95
Glu Asp Pro Tyr Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Arg
100 105 110
Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Arg Lys
115 120 125
Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr
130 135 140
Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser
145 150 155 160
Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr
165 170 175
Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys
180 185 190
His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro
195 200 205
Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 373
<211> 684
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA(A5H1EL1D) VH-CH1-VH-CH1 "EE" hu IgG1 Fc-впадина PGLALA
<400> 373
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Thr Asp Tyr
20 25 30
Tyr Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Phe Ile Gly Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Lys Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr
85 90 95
Tyr Cys Thr Arg Asp Arg Gly Leu Arg Phe Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Glu Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Glu Lys Val Glu Pro Lys Ser Cys
210 215 220
Asp Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Val Gln Leu Leu
225 230 235 240
Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser
245 250 255
Cys Ala Ala Ser Gly Phe Thr Phe Thr Asp Tyr Tyr Met Asn Trp Val
260 265 270
Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Leu Gly Phe Ile Gly Asn
275 280 285
Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala Ser Val Lys Gly Arg
290 295 300
Phe Thr Ile Ser Arg Asp Lys Ser Lys Asn Thr Leu Tyr Leu Gln Met
305 310 315 320
Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr Tyr Cys Thr Arg Asp
325 330 335
Arg Gly Leu Arg Phe Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Thr Val
340 345 350
Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala
355 360 365
Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu
370 375 380
Val Glu Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly
385 390 395 400
Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser
405 410 415
Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu
420 425 430
Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr
435 440 445
Lys Val Asp Glu Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr
450 455 460
Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe
465 470 475 480
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
485 490 495
Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val
500 505 510
Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
515 520 525
Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val
530 535 540
Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
545 550 555 560
Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr Ile Ser
565 570 575
Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Cys Thr Leu Pro Pro
580 585 590
Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Ser Cys Ala Val
595 600 605
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
610 615 620
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp
625 630 635 640
Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
645 650 655
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
660 665 670
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
675 680
<210> 374
<211> 672
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA) VL-CH1 CEA(A5H1EL1D) VH-CH1 "EE" hu IgG1 Fc-выступ
PGLALA
<400> 374
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys His Ala Ser Gln Asn Ile Tyr Val Trp
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gly Gln Thr Tyr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Ser Ser Ala Ser Thr
100 105 110
Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser
115 120 125
Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu
130 135 140
Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His
145 150 155 160
Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser
165 170 175
Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys
180 185 190
Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu
195 200 205
Pro Lys Ser Cys Asp Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu
210 215 220
Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser
225 230 235 240
Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Thr Asp Tyr Tyr
245 250 255
Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Leu Gly
260 265 270
Phe Ile Gly Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala Ser
275 280 285
Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Lys Ser Lys Asn Thr Leu
290 295 300
Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr Tyr
305 310 315 320
Cys Thr Arg Asp Arg Gly Leu Arg Phe Tyr Phe Asp Tyr Trp Gly Gln
325 330 335
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
340 345 350
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
355 360 365
Leu Gly Cys Leu Val Glu Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
370 375 380
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
385 390 395 400
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
405 410 415
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
420 425 430
Pro Ser Asn Thr Lys Val Asp Glu Lys Val Glu Pro Lys Ser Cys Asp
435 440 445
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
450 455 460
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
465 470 475 480
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
485 490 495
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
500 505 510
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
515 520 525
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
530 535 540
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
545 550 555 560
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
565 570 575
Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
580 585 590
Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
595 600 605
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
610 615 620
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
625 630 635 640
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
645 650 655
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
660 665 670
<210> 375
<211> 436
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA(P002.139) VL-CH1 hu IgG1 Fc-впадина PGLALA
<400> 375
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Ser Ser Val Thr Tyr Ile
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Ser Trp Ile Tyr
35 40 45
Ala Thr Ser Asn Leu Ala Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu
65 70 75 80
Asp Phe Ala Val Tyr Tyr Cys Gln His Trp Ser Ser Lys Pro Pro Thr
85 90 95
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Ser Ser Ala Ser Thr Lys
100 105 110
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
115 120 125
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
130 135 140
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
145 150 155 160
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
165 170 175
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
180 185 190
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro
195 200 205
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
210 215 220
Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
225 230 235 240
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
245 250 255
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
260 265 270
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
275 280 285
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
290 295 300
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly
305 310 315 320
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
325 330 335
Pro Gln Val Cys Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn
340 345 350
Gln Val Ser Leu Ser Cys Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile
355 360 365
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
370 375 380
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Val Ser Lys
385 390 395 400
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
405 410 415
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
420 425 430
Ser Leu Ser Pro
435
<210> 376
<211> 228
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA(P002.139) VH-C-каппа
<400> 376
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Thr Asp Tyr
20 25 30
Tyr Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Phe Ile Gly Asn Lys Ala Asn Ala Tyr Thr Thr Glu Tyr Ser Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Lys Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr
85 90 95
Tyr Cys Thr Arg Asp Arg Gly Leu Arg Phe Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Val Ala Ala Pro Ser
115 120 125
Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala
130 135 140
Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val
145 150 155 160
Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser
165 170 175
Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr
180 185 190
Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys
195 200 205
Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn
210 215 220
Arg Gly Glu Cys
225
<210> 377
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28 (SA_Variant 8) hu IgG1 легкая цепь
<400> 377
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys His Ala Ser Gln Asn Ile Tyr Val Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gly Gln Thr Tyr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 378
<211> 448
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA_Variant 8) hu IgG1 PGLALA тяжелая цепь
<400> 378
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Ser Ile Tyr Pro Gly Asn Val Gln Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Phe Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
<210> 379
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28 (SA_Variant 11) hu IgG1 легкая цепь
<400> 379
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys His Ala Ser Gln Asn Ile Tyr Val Trp
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gly Gln Thr Tyr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 380
<211> 448
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28 (SA_Variant 11) hu IgG1 PGLALA тяжелая цепь
<400> 380
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Ser Ile Tyr Pro Gly Asn Val Gln Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Phe Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
<210> 381
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28 (SA_Variant 15) hu IgG1 легкая цепь
<400> 381
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys His Ala Ser Gln Asn Ile Tyr Val Phe
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gly Gln Thr Tyr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 382
<211> 448
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28 (SA_Variant 15) hu IgG1 PGLALA тяжелая цепь
<400> 382
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Ser Ile Tyr Pro Gly Asn Val Gln Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Trp Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
<210> 383
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28 (SA_Variant 27) hu IgG1 легкая цепь
<400> 383
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys His Ala Ser Gln Gly Ile Tyr Val Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gly Gln Thr Tyr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 384
<211> 448
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(SA_Variant 27) hu IgG1 PGLALA тяжелая цепь
<400> 384
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Ser Ile Tyr Pro Gly Asn Val Asn Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Trp Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
<210> 385
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28 (SA_Variant 29) hu IgG1 легкая цепь
<400> 385
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys His Ala Ser Gln Asn Ile Tyr Val Trp
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gly Gln Thr Tyr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 386
<211> 448
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28 (SA_Variant 29) hu IgG1 PGLALA тяжелая цепь
<400> 386
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Ser Ile Tyr Pro Gly Asn Val Asn Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Trp Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
<210> 387
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> Avi tag
<400> 387
Gly Leu Asn Asp Ile Phe Glu Ala Gln Lys Ile Glu Trp His Glu
1 5 10 15
<210> 388
<211> 679
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28 VHCH1 "EE"-(G4S)2- FAP (4B9)-VHCH1 "EE" - Fc-выступ
PGLALA
<400> 388
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Cys Ile Tyr Pro Gly Asn Val Asn Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Arg Ala Thr Leu Thr Val Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Phe Cys
85 90 95
Thr Arg Ser His Tyr Gly Leu Asp Trp Asn Phe Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Glu Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Glu Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Val Gln Leu Leu Glu
225 230 235 240
Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys
245 250 255
Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr Ala Met Ser Trp Val Arg
260 265 270
Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ser Ala Ile Ile Gly Ser
275 280 285
Gly Ala Ser Thr Tyr Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile
290 295 300
Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu
305 310 315 320
Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala Lys Gly Trp Phe Gly
325 330 335
Gly Phe Asn Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala
340 345 350
Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser
355 360 365
Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Glu Asp Tyr Phe
370 375 380
Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly
385 390 395 400
Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu
405 410 415
Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr
420 425 430
Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Glu Lys
435 440 445
Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro
450 455 460
Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
465 470 475 480
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
485 490 495
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
500 505 510
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
515 520 525
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
530 535 540
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
545 550 555 560
Ala Leu Gly Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
565 570 575
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Cys Arg Asp Glu Leu
580 585 590
Thr Lys Asn Gln Val Ser Leu Trp Cys Leu Val Lys Gly Phe Tyr Pro
595 600 605
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
610 615 620
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
625 630 635 640
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
645 650 655
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
660 665 670
Lys Ser Leu Ser Leu Ser Pro
675
<210> 389
<211> 439
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28 VLCL "RK"-(G4S)2- FAP (4B9)-VLCL "RK"
<400> 389
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys His Ala Ser Gln Asn Ile Tyr Val Trp
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gly Gln Thr Tyr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Arg Lys Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
210 215 220
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
225 230 235 240
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Thr Ser Ser
245 250 255
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
260 265 270
Ile Asn Val Gly Ser Arg Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
275 280 285
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
290 295 300
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Gly Ile Met Leu Pro
305 310 315 320
Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala
325 330 335
Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Arg Lys Leu Lys Ser
340 345 350
Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu
355 360 365
Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser
370 375 380
Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu
385 390 395 400
Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val
405 410 415
Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys
420 425 430
Ser Phe Asn Arg Gly Glu Cys
435
<210> 390
<211> 455
<212> Белок
<213> Искусственная последовательность
<220>
<223> EpCAM (MT201) hu IgG1 VH-CH1 "EE" Fc-впадина PGLALA
<400> 390
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Ser Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Asp Met Gly Trp Gly Ser Gly Trp Arg Pro Tyr Tyr Tyr Tyr
100 105 110
Gly Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala
115 120 125
Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser
130 135 140
Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Glu Asp Tyr Phe
145 150 155 160
Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly
165 170 175
Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu
180 185 190
Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr
195 200 205
Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Glu Lys
210 215 220
Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro
225 230 235 240
Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
245 250 255
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
260 265 270
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
275 280 285
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
290 295 300
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
305 310 315 320
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
325 330 335
Ala Leu Gly Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
340 345 350
Pro Arg Glu Pro Gln Val Cys Thr Leu Pro Pro Ser Arg Asp Glu Leu
355 360 365
Thr Lys Asn Gln Val Ser Leu Ser Cys Ala Val Lys Gly Phe Tyr Pro
370 375 380
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
385 390 395 400
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
405 410 415
Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
420 425 430
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
435 440 445
Lys Ser Leu Ser Leu Ser Pro
450 455
<210> 391
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> EpCAM(MT201) VL-C-каппа "RK"
<400> 391
Glu Leu Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Thr Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro Lys Leu Leu Ile
35 40 45
Tyr Trp Ala Ser Thr Arg Glu Ser Gly Val Pro Asp Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ser Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Asp Ile Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Arg Lys Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 392
<211> 443
<212> Белок
<213> Искусственная последовательность
<220>
<223> HER3 VL-CH1 hu IgG1 Fc-впадина PGLALA
<400> 392
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Ile Asn Cys Lys Ser Ser Gln Ser Val Leu Asn Ser
20 25 30
Gly Asn Gln Lys Asn Tyr Leu Thr Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Pro Pro Lys Leu Leu Ile Tyr Trp Ala Ser Thr Arg Glu Ser Gly Val
50 55 60
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
65 70 75 80
Ile Ser Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Tyr Cys Gln Ser
85 90 95
Asp Tyr Ser Tyr Pro Tyr Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile
100 105 110
Lys Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro
115 120 125
Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val
130 135 140
Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala
145 150 155 160
Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly
165 170 175
Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly
180 185 190
Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys
195 200 205
Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys
210 215 220
Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu
225 230 235 240
Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu
245 250 255
Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys
260 265 270
Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys
275 280 285
Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu
290 295 300
Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys
305 310 315 320
Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr Ile Ser Lys
325 330 335
Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Cys Thr Leu Pro Pro Ser
340 345 350
Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Ser Cys Ala Val Lys
355 360 365
Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln
370 375 380
Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly
385 390 395 400
Ser Phe Phe Leu Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln
405 410 415
Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn
420 425 430
His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440
<210> 393
<211> 227
<212> Белок
<213> Искусственная последовательность
<220>
<223> HER3 VH-C-каппа
<400> 393
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Arg Ser Ser
20 25 30
Tyr Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Tyr Ala Gly Thr Gly Ser Pro Ser Tyr Asn Gln Lys Leu
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg His Arg Asp Tyr Tyr Ser Asn Ser Leu Thr Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Ala Ser Val Ala Ala Pro Ser Val
115 120 125
Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser
130 135 140
Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln
145 150 155 160
Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val
165 170 175
Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu
180 185 190
Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu
195 200 205
Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg
210 215 220
Gly Glu Cys
225
<210> 394
<211> 441
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD30 VL-CH1 hu IgG1 Fc-впадина PGLALA
<400> 394
Asp Ile Val Leu Thr Gln Ser Pro Ala Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Gln Arg Ala Thr Ile Ser Cys Lys Ala Ser Gln Ser Val Asp Phe Asp
20 25 30
Gly Asp Ser Tyr Met Asn Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro
35 40 45
Lys Val Leu Ile Tyr Ala Ala Ser Asn Leu Glu Ser Gly Ile Pro Ala
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Asn Ile His
65 70 75 80
Pro Val Glu Glu Glu Asp Ala Ala Thr Tyr Tyr Cys Gln Gln Ser Asn
85 90 95
Glu Asp Pro Trp Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Ser
100 105 110
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser
115 120 125
Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
130 135 140
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
145 150 155 160
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
165 170 175
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln
180 185 190
Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp
195 200 205
Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro
210 215 220
Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro
225 230 235 240
Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr
245 250 255
Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn
260 265 270
Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg
275 280 285
Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val
290 295 300
Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser
305 310 315 320
Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys
325 330 335
Gly Gln Pro Arg Glu Pro Gln Val Cys Thr Leu Pro Pro Ser Arg Asp
340 345 350
Glu Leu Thr Lys Asn Gln Val Ser Leu Ser Cys Ala Val Lys Gly Phe
355 360 365
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
370 375 380
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
385 390 395 400
Phe Leu Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly
405 410 415
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
420 425 430
Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440
<210> 395
<211> 224
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD30 VH-C-каппа
<400> 395
Gln Ile Gln Leu Gln Gln Ser Gly Pro Glu Val Val Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Ile Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Tyr Ile Thr Trp Val Lys Gln Lys Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Trp Ile Tyr Pro Gly Ser Gly Asn Thr Lys Tyr Asn Glu Lys Phe
50 55 60
Lys Gly Lys Ala Thr Leu Thr Val Asp Thr Ser Ser Ser Thr Ala Phe
65 70 75 80
Met Gln Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val Tyr Phe Cys
85 90 95
Ala Asn Tyr Gly Asn Tyr Trp Phe Ala Tyr Trp Gly Gln Gly Thr Gln
100 105 110
Val Thr Val Ser Ala Ala Ser Val Ala Ala Pro Ser Val Phe Ile Phe
115 120 125
Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys
130 135 140
Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val
145 150 155 160
Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln
165 170 175
Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser
180 185 190
Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His
195 200 205
Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215 220
<210> 396
<211> 437
<212> Белок
<213> Искусственная последовательность
<220>
<223> TPBG VL-CH1 hu IgG1 Fc-впадина PGLALA
<400> 396
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asp Ile Ser Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Gln Ile Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ala Asn Ser Phe Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Ser Ser Ala Ser Thr
100 105 110
Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser
115 120 125
Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu
130 135 140
Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His
145 150 155 160
Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser
165 170 175
Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys
180 185 190
Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu
195 200 205
Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro
210 215 220
Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
225 230 235 240
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
245 250 255
Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp
260 265 270
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr
275 280 285
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
290 295 300
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu
305 310 315 320
Gly Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
325 330 335
Glu Pro Gln Val Cys Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys
340 345 350
Asn Gln Val Ser Leu Ser Cys Ala Val Lys Gly Phe Tyr Pro Ser Asp
355 360 365
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
370 375 380
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Val Ser
385 390 395 400
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser
405 410 415
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
420 425 430
Leu Ser Leu Ser Pro
435
<210> 397
<211> 231
<212> Белок
<213> Искусственная последовательность
<220>
<223> TPBG VH-C-каппа
<400> 397
Glu Val His Leu Leu Glu Ser Gly Gly Gly Leu Val His Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Arg Ser Asp
20 25 30
Ala Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Val Ser Gly Ser Gly Gly Ser Pro Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Thr Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Gly Gly Ser Ile Ala Gly Ser Tyr Tyr Tyr Tyr Pro Met Asp
100 105 110
Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Val Ala
115 120 125
Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser
130 135 140
Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu
145 150 155 160
Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser
165 170 175
Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu
180 185 190
Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val
195 200 205
Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys
210 215 220
Ser Phe Asn Arg Gly Glu Cys
225 230
<210> 398
<211> 445
<212> Белок
<213> Искусственная последовательность
<220>
<223> GPRC5D (5E11) hu IgG1 VH-CH1 "EE" Fc-впадина PGLALA
<400> 398
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Lys Tyr
20 25 30
Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Ser Ile Ser Thr Gly Gly Val Asn Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr His Thr Gly Asp Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Met
100 105 110
Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu
115 120 125
Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys
130 135 140
Leu Val Glu Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser
145 150 155 160
Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser
165 170 175
Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser
180 185 190
Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn
195 200 205
Thr Lys Val Asp Glu Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His
210 215 220
Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val
225 230 235 240
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
245 250 255
Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu
260 265 270
Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
275 280 285
Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser
290 295 300
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
305 310 315 320
Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr Ile
325 330 335
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Cys Thr Leu Pro
340 345 350
Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Ser Cys Ala
355 360 365
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
370 375 380
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
385 390 395 400
Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val Asp Lys Ser Arg
405 410 415
Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
420 425 430
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
<210> 399
<211> 218
<212> Белок
<213> Искусственная последовательность
<220>
<223> GPRC5D (5E11)VL-C-каппа "RK"
<400> 399
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ile Ser
20 25 30
Gly Ile Asn Leu Met Asn Trp Tyr Gln Gln Lys Pro Gly Gln Gln Pro
35 40 45
Lys Leu Leu Ile Tyr His Ala Ser Ile Leu Ala Ser Gly Ile Pro Asp
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
65 70 75 80
Arg Leu Glu Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Thr Arg
85 90 95
Glu Ser Pro Leu Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys Arg
100 105 110
Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Arg Lys
115 120 125
Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr
130 135 140
Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser
145 150 155 160
Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr
165 170 175
Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys
180 185 190
His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro
195 200 205
Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 400
<211> 437
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD38 VL-CH1 hu IgG1 Fc-впадина PGLALA
<400> 400
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Arg Ala Thr Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Arg Ser Asn Trp Pro Pro
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Ser Ser Ala Ser Thr
100 105 110
Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser
115 120 125
Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu
130 135 140
Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His
145 150 155 160
Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser
165 170 175
Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys
180 185 190
Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu
195 200 205
Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro
210 215 220
Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
225 230 235 240
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
245 250 255
Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp
260 265 270
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr
275 280 285
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
290 295 300
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu
305 310 315 320
Gly Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
325 330 335
Glu Pro Gln Val Cys Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys
340 345 350
Asn Gln Val Ser Leu Ser Cys Ala Val Lys Gly Phe Tyr Pro Ser Asp
355 360 365
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
370 375 380
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Val Ser
385 390 395 400
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser
405 410 415
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
420 425 430
Leu Ser Leu Ser Pro
435
<210> 401
<211> 229
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD38 VH-C-каппа
<400> 401
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Val Ser Gly Phe Thr Phe Asn Ser Phe
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Gly Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Phe Cys
85 90 95
Ala Lys Asp Lys Ile Leu Trp Phe Gly Glu Pro Val Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Val Ala Ala Pro
115 120 125
Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr
130 135 140
Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys
145 150 155 160
Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu
165 170 175
Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser
180 185 190
Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala
195 200 205
Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe
210 215 220
Asn Arg Gly Glu Cys
225
<210> 402
<211> 443
<212> Белок
<213> Искусственная последовательность
<220>
<223> BCMA hu IgG1 VH-CH1 "EE" Fc-впадина PGLALA
<400> 402
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Thr Ala Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Tyr Trp Pro Met Ser Leu Trp Gly Gln Gly Thr Leu Val Thr
100 105 110
Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro
115 120 125
Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val
130 135 140
Glu Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala
145 150 155 160
Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly
165 170 175
Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly
180 185 190
Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys
195 200 205
Val Asp Glu Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys
210 215 220
Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu
225 230 235 240
Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu
245 250 255
Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys
260 265 270
Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys
275 280 285
Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu
290 295 300
Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys
305 310 315 320
Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr Ile Ser Lys
325 330 335
Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Cys Thr Leu Pro Pro Ser
340 345 350
Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Ser Cys Ala Val Lys
355 360 365
Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln
370 375 380
Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly
385 390 395 400
Ser Phe Phe Leu Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln
405 410 415
Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn
420 425 430
His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440
<210> 403
<211> 215
<212> Белок
<213> Искусственная последовательность
<220>
<223> BCMA VL-C-каппа "RK"
<400> 403
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ala Tyr
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Met Tyr Asp Ala Ser Ile Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Glu Arg Trp Pro
85 90 95
Leu Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala
100 105 110
Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Arg Lys Leu Lys Ser
115 120 125
Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu
130 135 140
Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser
145 150 155 160
Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu
165 170 175
Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val
180 185 190
Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys
195 200 205
Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 404
<211> 670
<212> Белок
<213> Искусственная последовательность
<220>
<223> GPRC5D (5E11) VH-CH1(EE)-CD3 VL-CH1-Fc (выступ, P329G LALA)
(GPRC5D
TCB)
<400> 404
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Lys Tyr
20 25 30
Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Ser Ile Ser Thr Gly Gly Val Asn Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr His Thr Gly Asp Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Met
100 105 110
Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu
115 120 125
Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys
130 135 140
Leu Val Glu Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser
145 150 155 160
Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser
165 170 175
Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser
180 185 190
Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn
195 200 205
Thr Lys Val Asp Glu Lys Val Glu Pro Lys Ser Cys Asp Gly Gly Gly
210 215 220
Gly Ser Gly Gly Gly Gly Ser Gln Ala Val Val Thr Gln Glu Pro Ser
225 230 235 240
Leu Thr Val Ser Pro Gly Gly Thr Val Thr Leu Thr Cys Gly Ser Ser
245 250 255
Thr Gly Ala Val Thr Thr Ser Asn Tyr Ala Asn Trp Val Gln Glu Lys
260 265 270
Pro Gly Gln Ala Phe Arg Gly Leu Ile Gly Gly Thr Asn Lys Arg Ala
275 280 285
Pro Gly Thr Pro Ala Arg Phe Ser Gly Ser Leu Leu Gly Gly Lys Ala
290 295 300
Ala Leu Thr Leu Ser Gly Ala Gln Pro Glu Asp Glu Ala Glu Tyr Tyr
305 310 315 320
Cys Ala Leu Trp Tyr Ser Asn Leu Trp Val Phe Gly Gly Gly Thr Lys
325 330 335
Leu Thr Val Leu Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
340 345 350
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
355 360 365
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
370 375 380
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
385 390 395 400
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
405 410 415
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
420 425 430
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
435 440 445
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser
450 455 460
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
465 470 475 480
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
485 490 495
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
500 505 510
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val
515 520 525
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
530 535 540
Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr
545 550 555 560
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
565 570 575
Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Trp Cys
580 585 590
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
595 600 605
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
610 615 620
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser
625 630 635 640
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
645 650 655
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
660 665 670
<210> 405
<211> 232
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD3 VH-CL (GPRC5D TCB)
<400> 405
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Gln Phe Ser Ser Tyr
20 25 30
Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Arg Ile Arg Ser Lys Tyr Asn Asn Tyr Ala Thr Tyr Tyr Ala Asp
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Val Arg His Thr Thr Phe Pro Ser Ser Tyr Val Ser Tyr Tyr
100 105 110
Gly Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Val
115 120 125
Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys
130 135 140
Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg
145 150 155 160
Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn
165 170 175
Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser
180 185 190
Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys
195 200 205
Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr
210 215 220
Lys Ser Phe Asn Arg Gly Glu Cys
225 230
<210> 406
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD19 (8B8-2B11) CDR-H1
<400> 406
Asp Tyr Ile Met His
1 5
<210> 407
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD19 (8B8-2B11) CDR-H2
<400> 407
Tyr Ile Asn Pro Tyr Asn Asp Gly Ser Lys Tyr Thr Glu Lys Phe Gln
1 5 10 15
Gly
<210> 408
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD19 (8B8-2B11) CDR-H3
<400> 408
Gly Thr Tyr Tyr Tyr Gly Pro Gln Leu Phe Asp Tyr
1 5 10
<210> 409
<211> 16
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD19 (8B8-2B11) CDR-L1
<400> 409
Lys Ser Ser Gln Ser Leu Glu Thr Ser Thr Gly Thr Thr Tyr Leu Asn
1 5 10 15
<210> 410
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD19 (8B8-2B11) CDR-L2
<400> 410
Arg Val Ser Lys Arg Phe Ser
1 5
<210> 411
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD19 (8B8-2B11) CDR-L3
<400> 411
Leu Gln Leu Leu Glu Asp Pro Tyr Thr
1 5
<210> 412
<211> 121
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD19 (8B8-2B11) VH
<400> 412
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Ile Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Tyr Ile Asn Pro Tyr Asn Asp Gly Ser Lys Tyr Thr Glu Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Ser Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Thr Tyr Tyr Tyr Gly Pro Gln Leu Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 413
<211> 112
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD19 (8B8-2B11) VL
<400> 413
Asp Ile Val Met Thr Gln Thr Pro Leu Ser Leu Ser Val Thr Pro Gly
1 5 10 15
Gln Pro Ala Ser Ile Ser Cys Lys Ser Ser Gln Ser Leu Glu Thr Ser
20 25 30
Thr Gly Thr Thr Tyr Leu Asn Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Arg Val Ser Lys Arg Phe Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Leu Gln Leu
85 90 95
Leu Glu Asp Pro Tyr Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105 110
<210> 414
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD19 (8B8-018) CDR-H1
<400> 414
Asp Tyr Ile Met His
1 5
<210> 415
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD19 (8B8-018) CDR-H2
<400> 415
Tyr Ile Asn Pro Tyr Asn Asp Gly Ser Lys Tyr Thr Glu Lys Phe Gln
1 5 10 15
Gly
<210> 416
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD19 (8B8-018) CDR-H3
<400> 416
Gly Thr Tyr Tyr Tyr Gly Ser Ala Leu Phe Asp Tyr
1 5 10
<210> 417
<211> 16
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD19 (8B8-018) CDR-L1
<400> 417
Lys Ser Ser Gln Ser Leu Glu Asn Pro Asn Gly Asn Thr Tyr Leu Asn
1 5 10 15
<210> 418
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD19 (8B8-018) CDR-L2
<400> 418
Arg Val Ser Lys Arg Phe Ser
1 5
<210> 419
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD19 (8B8-018) CDR-L3
<400> 419
Leu Gln Leu Thr His Val Pro Tyr Thr
1 5
<210> 420
<211> 121
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD19 (8B8-018) VH
<400> 420
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Ile Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Tyr Ile Asn Pro Tyr Asn Asp Gly Ser Lys Tyr Thr Glu Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Ser Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Thr Tyr Tyr Tyr Gly Ser Ala Leu Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 421
<211> 112
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD19 (8B8-018) VL
<400> 421
Asp Ile Val Met Thr Gln Thr Pro Leu Ser Leu Ser Val Thr Pro Gly
1 5 10 15
Gln Pro Ala Ser Ile Ser Cys Lys Ser Ser Gln Ser Leu Glu Asn Pro
20 25 30
Asn Gly Asn Thr Tyr Leu Asn Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Arg Val Ser Lys Arg Phe Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Leu Gln Leu
85 90 95
Thr His Val Pro Tyr Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105 110
<210> 422
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD79b (huMA79b.v28) CDR-H1
<400> 422
Ser Tyr Trp Ile Glu
1 5
<210> 423
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD79b (huMA79b.v28) CDR-H2
<400> 423
Glu Ile Leu Pro Gly Gly Gly Asp Thr Asn Tyr Asn Glu Ile Phe Lys
1 5 10 15
Gly
<210> 424
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD79b (huMA79b.v28) CDR-H3
<400> 424
Arg Val Pro Ile Arg Leu Asp Tyr
1 5
<210> 425
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD79b (huMA79b.v28) CDR-L1
<400> 425
Lys Ala Ser Gln Ser Val Asp Tyr Glu Gly Asp Ser Phe Leu Asn
1 5 10 15
<210> 426
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD79b (huMA79b.v28) CDR-L2
<400> 426
Ala Ala Ser Asn Leu Glu Ser
1 5
<210> 427
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD79b (huMA79b.v28) CDR-L3
<400> 427
Gln Gln Ser Asn Glu Asp Pro Leu Thr
1 5
<210> 428
<211> 117
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD79b (huMA79b.v28) VH
<400> 428
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Tyr Thr Phe Ser Ser Tyr
20 25 30
Trp Ile Glu Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Glu Ile Leu Pro Gly Gly Gly Asp Thr Asn Tyr Asn Glu Ile Phe
50 55 60
Lys Gly Arg Ala Thr Phe Ser Ala Asp Thr Ser Lys Asn Thr Ala Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Arg Arg Val Pro Ile Arg Leu Asp Tyr Trp Gly Gln Gly Thr Leu
100 105 110
Val Thr Val Ser Ser
115
<210> 429
<211> 111
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD79b (huMA79b.v28) VL
<400> 429
Asp Ile Gln Leu Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Ser Val Asp Tyr Glu
20 25 30
Gly Asp Ser Phe Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro
35 40 45
Lys Leu Leu Ile Tyr Ala Ala Ser Asn Leu Glu Ser Gly Val Pro Ser
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
65 70 75 80
Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Asn
85 90 95
Glu Asp Pro Leu Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105 110
<210> 430
<211> 442
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL (CD19 2B11) -CH1 Fc-впадина PGLALA
<400> 430
Asp Ile Val Met Thr Gln Thr Pro Leu Ser Leu Ser Val Thr Pro Gly
1 5 10 15
Gln Pro Ala Ser Ile Ser Cys Lys Ser Ser Gln Ser Leu Glu Thr Ser
20 25 30
Thr Gly Thr Thr Tyr Leu Asn Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Arg Val Ser Lys Arg Phe Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Leu Gln Leu
85 90 95
Leu Glu Asp Pro Tyr Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105 110
Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser
115 120 125
Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys
130 135 140
Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu
145 150 155 160
Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu
165 170 175
Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr
180 185 190
Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val
195 200 205
Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro
210 215 220
Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe
225 230 235 240
Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val
245 250 255
Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe
260 265 270
Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro
275 280 285
Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr
290 295 300
Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val
305 310 315 320
Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala
325 330 335
Lys Gly Gln Pro Arg Glu Pro Gln Val Cys Thr Leu Pro Pro Ser Arg
340 345 350
Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Ser Cys Ala Val Lys Gly
355 360 365
Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro
370 375 380
Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser
385 390 395 400
Phe Phe Leu Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln
405 410 415
Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His
420 425 430
Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440
<210> 431
<211> 228
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH (CD19 2B11) CL
<400> 431
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Ile Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Tyr Ile Asn Pro Tyr Asn Asp Gly Ser Lys Tyr Thr Glu Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Ser Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Thr Tyr Tyr Tyr Gly Pro Gln Leu Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Val Ala Ala Pro Ser
115 120 125
Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala
130 135 140
Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val
145 150 155 160
Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser
165 170 175
Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr
180 185 190
Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys
195 200 205
Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn
210 215 220
Arg Gly Glu Cys
225
<210> 432
<211> 441
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL (huMA79b.v28) -CH1 Fc-впадина PGLALA
<400> 432
Asp Ile Gln Leu Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Ser Val Asp Tyr Glu
20 25 30
Gly Asp Ser Phe Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro
35 40 45
Lys Leu Leu Ile Tyr Ala Ala Ser Asn Leu Glu Ser Gly Val Pro Ser
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
65 70 75 80
Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Asn
85 90 95
Glu Asp Pro Leu Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Ser
100 105 110
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser
115 120 125
Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
130 135 140
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
145 150 155 160
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
165 170 175
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln
180 185 190
Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp
195 200 205
Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro
210 215 220
Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro
225 230 235 240
Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr
245 250 255
Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn
260 265 270
Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg
275 280 285
Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val
290 295 300
Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser
305 310 315 320
Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys
325 330 335
Gly Gln Pro Arg Glu Pro Gln Val Cys Thr Leu Pro Pro Ser Arg Asp
340 345 350
Glu Leu Thr Lys Asn Gln Val Ser Leu Ser Cys Ala Val Lys Gly Phe
355 360 365
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
370 375 380
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
385 390 395 400
Phe Leu Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly
405 410 415
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
420 425 430
Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440
<210> 433
<211> 224
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH (huMA79b.v28) CL
<400> 433
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Tyr Thr Phe Ser Ser Tyr
20 25 30
Trp Ile Glu Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Glu Ile Leu Pro Gly Gly Gly Asp Thr Asn Tyr Asn Glu Ile Phe
50 55 60
Lys Gly Arg Ala Thr Phe Ser Ala Asp Thr Ser Lys Asn Thr Ala Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Arg Arg Val Pro Ile Arg Leu Asp Tyr Trp Gly Gln Gly Thr Leu
100 105 110
Val Thr Val Ser Ser Ala Ser Val Ala Ala Pro Ser Val Phe Ile Phe
115 120 125
Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys
130 135 140
Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val
145 150 155 160
Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln
165 170 175
Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser
180 185 190
Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His
195 200 205
Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215 220
<210> 434
<211> 556
<212> Белок
<213> Homo sapiens
<400> 434
Met Pro Pro Pro Arg Leu Leu Phe Phe Leu Leu Phe Leu Thr Pro Met
1 5 10 15
Glu Val Arg Pro Glu Glu Pro Leu Val Val Lys Val Glu Glu Gly Asp
20 25 30
Asn Ala Val Leu Gln Cys Leu Lys Gly Thr Ser Asp Gly Pro Thr Gln
35 40 45
Gln Leu Thr Trp Ser Arg Glu Ser Pro Leu Lys Pro Phe Leu Lys Leu
50 55 60
Ser Leu Gly Leu Pro Gly Leu Gly Ile His Met Arg Pro Leu Ala Ile
65 70 75 80
Trp Leu Phe Ile Phe Asn Val Ser Gln Gln Met Gly Gly Phe Tyr Leu
85 90 95
Cys Gln Pro Gly Pro Pro Ser Glu Lys Ala Trp Gln Pro Gly Trp Thr
100 105 110
Val Asn Val Glu Gly Ser Gly Glu Leu Phe Arg Trp Asn Val Ser Asp
115 120 125
Leu Gly Gly Leu Gly Cys Gly Leu Lys Asn Arg Ser Ser Glu Gly Pro
130 135 140
Ser Ser Pro Ser Gly Lys Leu Met Ser Pro Lys Leu Tyr Val Trp Ala
145 150 155 160
Lys Asp Arg Pro Glu Ile Trp Glu Gly Glu Pro Pro Cys Leu Pro Pro
165 170 175
Arg Asp Ser Leu Asn Gln Ser Leu Ser Gln Asp Leu Thr Met Ala Pro
180 185 190
Gly Ser Thr Leu Trp Leu Ser Cys Gly Val Pro Pro Asp Ser Val Ser
195 200 205
Arg Gly Pro Leu Ser Trp Thr His Val His Pro Lys Gly Pro Lys Ser
210 215 220
Leu Leu Ser Leu Glu Leu Lys Asp Asp Arg Pro Ala Arg Asp Met Trp
225 230 235 240
Val Met Glu Thr Gly Leu Leu Leu Pro Arg Ala Thr Ala Gln Asp Ala
245 250 255
Gly Lys Tyr Tyr Cys His Arg Gly Asn Leu Thr Met Ser Phe His Leu
260 265 270
Glu Ile Thr Ala Arg Pro Val Leu Trp His Trp Leu Leu Arg Thr Gly
275 280 285
Gly Trp Lys Val Ser Ala Val Thr Leu Ala Tyr Leu Ile Phe Cys Leu
290 295 300
Cys Ser Leu Val Gly Ile Leu His Leu Gln Arg Ala Leu Val Leu Arg
305 310 315 320
Arg Lys Arg Lys Arg Met Thr Asp Pro Thr Arg Arg Phe Phe Lys Val
325 330 335
Thr Pro Pro Pro Gly Ser Gly Pro Gln Asn Gln Tyr Gly Asn Val Leu
340 345 350
Ser Leu Pro Thr Pro Thr Ser Gly Leu Gly Arg Ala Gln Arg Trp Ala
355 360 365
Ala Gly Leu Gly Gly Thr Ala Pro Ser Tyr Gly Asn Pro Ser Ser Asp
370 375 380
Val Gln Ala Asp Gly Ala Leu Gly Ser Arg Ser Pro Pro Gly Val Gly
385 390 395 400
Pro Glu Glu Glu Glu Gly Glu Gly Tyr Glu Glu Pro Asp Ser Glu Glu
405 410 415
Asp Ser Glu Phe Tyr Glu Asn Asp Ser Asn Leu Gly Gln Asp Gln Leu
420 425 430
Ser Gln Asp Gly Ser Gly Tyr Glu Asn Pro Glu Asp Glu Pro Leu Gly
435 440 445
Pro Glu Asp Glu Asp Ser Phe Ser Asn Ala Glu Ser Tyr Glu Asn Glu
450 455 460
Asp Glu Glu Leu Thr Gln Pro Val Ala Arg Thr Met Asp Phe Leu Ser
465 470 475 480
Pro His Gly Ser Ala Trp Asp Pro Ser Arg Glu Ala Thr Ser Leu Gly
485 490 495
Ser Gln Ser Tyr Glu Asp Met Arg Gly Ile Leu Tyr Ala Ala Pro Gln
500 505 510
Leu Arg Ser Ile Arg Gly Gln Pro Gly Pro Asn His Glu Glu Asp Ala
515 520 525
Asp Ser Tyr Glu Asn Met Asp Asn Pro Asp Gly Pro Asp Pro Ala Trp
530 535 540
Gly Gly Gly Gly Arg Met Gly Thr Trp Ser Thr Arg
545 550 555
<210> 435
<211> 229
<212> Белок
<213> Homo sapiens
<400> 435
Met Ala Arg Leu Ala Leu Ser Pro Val Pro Ser His Trp Met Val Ala
1 5 10 15
Leu Leu Leu Leu Leu Ser Ala Glu Pro Val Pro Ala Ala Arg Ser Glu
20 25 30
Asp Arg Tyr Arg Asn Pro Lys Gly Ser Ala Cys Ser Arg Ile Trp Gln
35 40 45
Ser Pro Arg Phe Ile Ala Arg Lys Arg Gly Phe Thr Val Lys Met His
50 55 60
Cys Tyr Met Asn Ser Ala Ser Gly Asn Val Ser Trp Leu Trp Lys Gln
65 70 75 80
Glu Met Asp Glu Asn Pro Gln Gln Leu Lys Leu Glu Lys Gly Arg Met
85 90 95
Glu Glu Ser Gln Asn Glu Ser Leu Ala Thr Leu Thr Ile Gln Gly Ile
100 105 110
Arg Phe Glu Asp Asn Gly Ile Tyr Phe Cys Gln Gln Lys Cys Asn Asn
115 120 125
Thr Ser Glu Val Tyr Gln Gly Cys Gly Thr Glu Leu Arg Val Met Gly
130 135 140
Phe Ser Thr Leu Ala Gln Leu Lys Gln Arg Asn Thr Leu Lys Asp Gly
145 150 155 160
Ile Ile Met Ile Gln Thr Leu Leu Ile Ile Leu Phe Ile Ile Val Pro
165 170 175
Ile Phe Leu Leu Leu Asp Lys Asp Asp Ser Lys Ala Gly Met Glu Glu
180 185 190
Asp His Thr Tyr Glu Gly Leu Asp Ile Asp Gln Thr Ala Thr Tyr Glu
195 200 205
Asp Ile Val Thr Leu Arg Thr Gly Glu Val Lys Trp Ser Val Gly Glu
210 215 220
His Pro Gly Gln Glu
225
<210> 436
<211> 297
<212> Белок
<213> Homo sapiens
<400> 436
Met Thr Thr Pro Arg Asn Ser Val Asn Gly Thr Phe Pro Ala Glu Pro
1 5 10 15
Met Lys Gly Pro Ile Ala Met Gln Ser Gly Pro Lys Pro Leu Phe Arg
20 25 30
Arg Met Ser Ser Leu Val Gly Pro Thr Gln Ser Phe Phe Met Arg Glu
35 40 45
Ser Lys Thr Leu Gly Ala Val Gln Ile Met Asn Gly Leu Phe His Ile
50 55 60
Ala Leu Gly Gly Leu Leu Met Ile Pro Ala Gly Ile Tyr Ala Pro Ile
65 70 75 80
Cys Val Thr Val Trp Tyr Pro Leu Trp Gly Gly Ile Met Tyr Ile Ile
85 90 95
Ser Gly Ser Leu Leu Ala Ala Thr Glu Lys Asn Ser Arg Lys Cys Leu
100 105 110
Val Lys Gly Lys Met Ile Met Asn Ser Leu Ser Leu Phe Ala Ala Ile
115 120 125
Ser Gly Met Ile Leu Ser Ile Met Asp Ile Leu Asn Ile Lys Ile Ser
130 135 140
His Phe Leu Lys Met Glu Ser Leu Asn Phe Ile Arg Ala His Thr Pro
145 150 155 160
Tyr Ile Asn Ile Tyr Asn Cys Glu Pro Ala Asn Pro Ser Glu Lys Asn
165 170 175
Ser Pro Ser Thr Gln Tyr Cys Tyr Ser Ile Gln Ser Leu Phe Leu Gly
180 185 190
Ile Leu Ser Val Met Leu Ile Phe Ala Phe Phe Gln Glu Leu Val Ile
195 200 205
Ala Gly Ile Val Glu Asn Glu Trp Lys Arg Thr Cys Ser Arg Pro Lys
210 215 220
Ser Asn Ile Val Leu Leu Ser Ala Glu Glu Lys Lys Glu Gln Thr Ile
225 230 235 240
Glu Ile Lys Glu Glu Val Val Gly Leu Thr Glu Thr Ser Ser Gln Pro
245 250 255
Lys Asn Glu Glu Asp Ile Glu Ile Ile Pro Ile Gln Glu Glu Glu Glu
260 265 270
Glu Glu Thr Glu Thr Asn Phe Pro Glu Pro Pro Gln Asp Gln Glu Ser
275 280 285
Ser Pro Ile Glu Asn Asp Ser Ser Pro
290 295
<210> 437
<211> 847
<212> Белок
<213> Homo sapiens
<400> 437
Met His Leu Leu Gly Pro Trp Leu Leu Leu Leu Val Leu Glu Tyr Leu
1 5 10 15
Ala Phe Ser Asp Ser Ser Lys Trp Val Phe Glu His Pro Glu Thr Leu
20 25 30
Tyr Ala Trp Glu Gly Ala Cys Val Trp Ile Pro Cys Thr Tyr Arg Ala
35 40 45
Leu Asp Gly Asp Leu Glu Ser Phe Ile Leu Phe His Asn Pro Glu Tyr
50 55 60
Asn Lys Asn Thr Ser Lys Phe Asp Gly Thr Arg Leu Tyr Glu Ser Thr
65 70 75 80
Lys Asp Gly Lys Val Pro Ser Glu Gln Lys Arg Val Gln Phe Leu Gly
85 90 95
Asp Lys Asn Lys Asn Cys Thr Leu Ser Ile His Pro Val His Leu Asn
100 105 110
Asp Ser Gly Gln Leu Gly Leu Arg Met Glu Ser Lys Thr Glu Lys Trp
115 120 125
Met Glu Arg Ile His Leu Asn Val Ser Glu Arg Pro Phe Pro Pro His
130 135 140
Ile Gln Leu Pro Pro Glu Ile Gln Glu Ser Gln Glu Val Thr Leu Thr
145 150 155 160
Cys Leu Leu Asn Phe Ser Cys Tyr Gly Tyr Pro Ile Gln Leu Gln Trp
165 170 175
Leu Leu Glu Gly Val Pro Met Arg Gln Ala Ala Val Thr Ser Thr Ser
180 185 190
Leu Thr Ile Lys Ser Val Phe Thr Arg Ser Glu Leu Lys Phe Ser Pro
195 200 205
Gln Trp Ser His His Gly Lys Ile Val Thr Cys Gln Leu Gln Asp Ala
210 215 220
Asp Gly Lys Phe Leu Ser Asn Asp Thr Val Gln Leu Asn Val Lys His
225 230 235 240
Thr Pro Lys Leu Glu Ile Lys Val Thr Pro Ser Asp Ala Ile Val Arg
245 250 255
Glu Gly Asp Ser Val Thr Met Thr Cys Glu Val Ser Ser Ser Asn Pro
260 265 270
Glu Tyr Thr Thr Val Ser Trp Leu Lys Asp Gly Thr Ser Leu Lys Lys
275 280 285
Gln Asn Thr Phe Thr Leu Asn Leu Arg Glu Val Thr Lys Asp Gln Ser
290 295 300
Gly Lys Tyr Cys Cys Gln Val Ser Asn Asp Val Gly Pro Gly Arg Ser
305 310 315 320
Glu Glu Val Phe Leu Gln Val Gln Tyr Ala Pro Glu Pro Ser Thr Val
325 330 335
Gln Ile Leu His Ser Pro Ala Val Glu Gly Ser Gln Val Glu Phe Leu
340 345 350
Cys Met Ser Leu Ala Asn Pro Leu Pro Thr Asn Tyr Thr Trp Tyr His
355 360 365
Asn Gly Lys Glu Met Gln Gly Arg Thr Glu Glu Lys Val His Ile Pro
370 375 380
Lys Ile Leu Pro Trp His Ala Gly Thr Tyr Ser Cys Val Ala Glu Asn
385 390 395 400
Ile Leu Gly Thr Gly Gln Arg Gly Pro Gly Ala Glu Leu Asp Val Gln
405 410 415
Tyr Pro Pro Lys Lys Val Thr Thr Val Ile Gln Asn Pro Met Pro Ile
420 425 430
Arg Glu Gly Asp Thr Val Thr Leu Ser Cys Asn Tyr Asn Ser Ser Asn
435 440 445
Pro Ser Val Thr Arg Tyr Glu Trp Lys Pro His Gly Ala Trp Glu Glu
450 455 460
Pro Ser Leu Gly Val Leu Lys Ile Gln Asn Val Gly Trp Asp Asn Thr
465 470 475 480
Thr Ile Ala Cys Ala Ala Cys Asn Ser Trp Cys Ser Trp Ala Ser Pro
485 490 495
Val Ala Leu Asn Val Gln Tyr Ala Pro Arg Asp Val Arg Val Arg Lys
500 505 510
Ile Lys Pro Leu Ser Glu Ile His Ser Gly Asn Ser Val Ser Leu Gln
515 520 525
Cys Asp Phe Ser Ser Ser His Pro Lys Glu Val Gln Phe Phe Trp Glu
530 535 540
Lys Asn Gly Arg Leu Leu Gly Lys Glu Ser Gln Leu Asn Phe Asp Ser
545 550 555 560
Ile Ser Pro Glu Asp Ala Gly Ser Tyr Ser Cys Trp Val Asn Asn Ser
565 570 575
Ile Gly Gln Thr Ala Ser Lys Ala Trp Thr Leu Glu Val Leu Tyr Ala
580 585 590
Pro Arg Arg Leu Arg Val Ser Met Ser Pro Gly Asp Gln Val Met Glu
595 600 605
Gly Lys Ser Ala Thr Leu Thr Cys Glu Ser Asp Ala Asn Pro Pro Val
610 615 620
Ser His Tyr Thr Trp Phe Asp Trp Asn Asn Gln Ser Leu Pro Tyr His
625 630 635 640
Ser Gln Lys Leu Arg Leu Glu Pro Val Lys Val Gln His Ser Gly Ala
645 650 655
Tyr Trp Cys Gln Gly Thr Asn Ser Val Gly Lys Gly Arg Ser Pro Leu
660 665 670
Ser Thr Leu Thr Val Tyr Tyr Ser Pro Glu Thr Ile Gly Arg Arg Val
675 680 685
Ala Val Gly Leu Gly Ser Cys Leu Ala Ile Leu Ile Leu Ala Ile Cys
690 695 700
Gly Leu Lys Leu Gln Arg Arg Trp Lys Arg Thr Gln Ser Gln Gln Gly
705 710 715 720
Leu Gln Glu Asn Ser Ser Gly Gln Ser Phe Phe Val Arg Asn Lys Lys
725 730 735
Val Arg Arg Ala Pro Leu Ser Glu Gly Pro His Ser Leu Gly Cys Tyr
740 745 750
Asn Pro Met Met Glu Asp Gly Ile Ser Tyr Thr Thr Leu Arg Phe Pro
755 760 765
Glu Met Asn Ile Pro Arg Thr Gly Asp Ala Glu Ser Ser Glu Met Gln
770 775 780
Arg Pro Pro Pro Asp Cys Asp Asp Thr Val Thr Tyr Ser Ala Leu His
785 790 795 800
Lys Arg Gln Val Gly Asp Tyr Glu Asn Val Ile Pro Asp Phe Pro Glu
805 810 815
Asp Glu Gly Ile His Tyr Ser Glu Leu Ile Gln Phe Gly Val Gly Glu
820 825 830
Arg Pro Gln Ala Gln Glu Asn Val Asp Tyr Val Ile Leu Lys His
835 840 845
<210> 438
<211> 281
<212> Белок
<213> Homo sapiens
<400> 438
Met Ser Ala Gln Glu Ser Cys Leu Ser Leu Ile Lys Tyr Phe Leu Phe
1 5 10 15
Val Phe Asn Leu Phe Phe Phe Val Leu Gly Ser Leu Ile Phe Cys Phe
20 25 30
Gly Ile Trp Ile Leu Ile Asp Lys Thr Ser Phe Val Ser Phe Val Gly
35 40 45
Leu Ala Phe Val Pro Leu Gln Ile Trp Ser Lys Val Leu Ala Ile Ser
50 55 60
Gly Ile Phe Thr Met Gly Ile Ala Leu Leu Gly Cys Val Gly Ala Leu
65 70 75 80
Lys Glu Leu Arg Cys Leu Leu Gly Leu Tyr Phe Gly Met Leu Leu Leu
85 90 95
Leu Phe Ala Thr Gln Ile Thr Leu Gly Ile Leu Ile Ser Thr Gln Arg
100 105 110
Ala Gln Leu Glu Arg Ser Leu Arg Asp Val Val Glu Lys Thr Ile Gln
115 120 125
Lys Tyr Gly Thr Asn Pro Glu Glu Thr Ala Ala Glu Glu Ser Trp Asp
130 135 140
Tyr Val Gln Phe Gln Leu Arg Cys Cys Gly Trp His Tyr Pro Gln Asp
145 150 155 160
Trp Phe Gln Val Leu Ile Leu Arg Gly Asn Gly Ser Glu Ala His Arg
165 170 175
Val Pro Cys Ser Cys Tyr Asn Leu Ser Ala Thr Asn Asp Ser Thr Ile
180 185 190
Leu Asp Lys Val Ile Leu Pro Gln Leu Ser Arg Leu Gly His Leu Ala
195 200 205
Arg Ser Arg His Ser Ala Asp Ile Cys Ala Val Pro Ala Glu Ser His
210 215 220
Ile Tyr Arg Glu Gly Cys Ala Gln Gly Leu Gln Lys Trp Leu His Asn
225 230 235 240
Asn Leu Ile Ser Ile Val Gly Ile Cys Leu Gly Val Gly Leu Leu Glu
245 250 255
Leu Gly Phe Met Thr Leu Ser Ile Phe Leu Cys Arg Asn Leu Asp His
260 265 270
Val Tyr Asn Arg Leu Ala Arg Tyr Arg
275 280
<210> 439
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD3-HCDR1
<400> 439
Thr Tyr Ala Met Asn
1 5
<210> 440
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD3-HCDR2
<400> 440
Arg Ile Arg Ser Lys Tyr Asn Asn Tyr Ala Thr Tyr Tyr Ala Asp Ser
1 5 10 15
Val Lys Gly
<210> 441
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD3-HCDR3
<400> 441
His Gly Asn Phe Gly Asn Ser Tyr Val Ser Trp Phe Ala Tyr
1 5 10
<210> 442
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD3-LCDR1
<400> 442
Gly Ser Ser Thr Gly Ala Val Thr Thr Ser Asn Tyr Ala Asn
1 5 10
<210> 443
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD3-LCDR2
<400> 443
Gly Thr Asn Lys Arg Ala Pro
1 5
<210> 444
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD3-LCDR3
<400> 444
Ala Leu Trp Tyr Ser Asn Leu Trp Val
1 5
<210> 445
<211> 125
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD3 VH
<400> 445
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Thr Tyr
20 25 30
Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Arg Ile Arg Ser Lys Tyr Asn Asn Tyr Ala Thr Tyr Tyr Ala Asp
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Val Arg His Gly Asn Phe Gly Asn Ser Tyr Val Ser Trp Phe
100 105 110
Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 446
<211> 109
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD3 VL
<400> 446
Gln Ala Val Val Thr Gln Glu Pro Ser Leu Thr Val Ser Pro Gly Gly
1 5 10 15
Thr Val Thr Leu Thr Cys Gly Ser Ser Thr Gly Ala Val Thr Thr Ser
20 25 30
Asn Tyr Ala Asn Trp Val Gln Glu Lys Pro Gly Gln Ala Phe Arg Gly
35 40 45
Leu Ile Gly Gly Thr Asn Lys Arg Ala Pro Gly Thr Pro Ala Arg Phe
50 55 60
Ser Gly Ser Leu Leu Gly Gly Lys Ala Ala Leu Thr Leu Ser Gly Ala
65 70 75 80
Gln Pro Glu Asp Glu Ala Glu Tyr Tyr Cys Ala Leu Trp Tyr Ser Asn
85 90 95
Leu Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105
<210> 447
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD20-HCDR1
<400> 447
Tyr Ser Trp Ile Asn
1 5
<210> 448
<211> 16
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD20-HCDR2
<400> 448
Arg Ile Phe Pro Gly Asp Gly Asp Thr Asp Tyr Asn Gly Lys Phe Lys
1 5 10 15
<210> 449
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD20-HCDR3
<400> 449
Asn Val Phe Asp Gly Tyr Trp Leu Val Tyr
1 5 10
<210> 450
<211> 16
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD20-LCDR1
<400> 450
Arg Ser Ser Lys Ser Leu Leu His Ser Asn Gly Ile Thr Tyr Leu Tyr
1 5 10 15
<210> 451
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD20-LCDR2
<400> 451
Gln Met Ser Asn Leu Val Ser
1 5
<210> 452
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD20-LCDR3
<400> 452
Ala Gln Asn Leu Glu Leu Pro Tyr Thr
1 5
<210> 453
<211> 119
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD20 VH
<400> 453
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Ala Phe Ser Tyr Ser
20 25 30
Trp Ile Asn Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Arg Ile Phe Pro Gly Asp Gly Asp Thr Asp Tyr Asn Gly Lys Phe
50 55 60
Lys Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asn Val Phe Asp Gly Tyr Trp Leu Val Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 454
<211> 115
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD20 VL
<400> 454
Asp Ile Val Met Thr Gln Thr Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Lys Ser Leu Leu His Ser
20 25 30
Asn Gly Ile Thr Tyr Leu Tyr Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Gln Met Ser Asn Leu Val Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Ala Gln Asn
85 90 95
Leu Glu Leu Pro Tyr Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105 110
Arg Thr Val
115
<210> 455
<211> 672
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD20 VH-CH1(EE)-CD3 VL-CH1-Fc (выступ, P329G LALA)CD20
VH-CH1(EE)-CD3 VL-CH1-Fc (выступ, P329G LALA)
<400> 455
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Ala Phe Ser Tyr Ser
20 25 30
Trp Ile Asn Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Arg Ile Phe Pro Gly Asp Gly Asp Thr Asp Tyr Asn Gly Lys Phe
50 55 60
Lys Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asn Val Phe Asp Gly Tyr Trp Leu Val Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe
115 120 125
Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu
130 135 140
Gly Cys Leu Val Glu Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
145 150 155 160
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
165 170 175
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
180 185 190
Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro
195 200 205
Ser Asn Thr Lys Val Asp Glu Lys Val Glu Pro Lys Ser Cys Asp Gly
210 215 220
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ala Val Val Thr Gln Glu
225 230 235 240
Pro Ser Leu Thr Val Ser Pro Gly Gly Thr Val Thr Leu Thr Cys Gly
245 250 255
Ser Ser Thr Gly Ala Val Thr Thr Ser Asn Tyr Ala Asn Trp Val Gln
260 265 270
Glu Lys Pro Gly Gln Ala Phe Arg Gly Leu Ile Gly Gly Thr Asn Lys
275 280 285
Arg Ala Pro Gly Thr Pro Ala Arg Phe Ser Gly Ser Leu Leu Gly Gly
290 295 300
Lys Ala Ala Leu Thr Leu Ser Gly Ala Gln Pro Glu Asp Glu Ala Glu
305 310 315 320
Tyr Tyr Cys Ala Leu Trp Tyr Ser Asn Leu Trp Val Phe Gly Gly Gly
325 330 335
Thr Lys Leu Thr Val Leu Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
340 345 350
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
355 360 365
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
370 375 380
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
385 390 395 400
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
405 410 415
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
420 425 430
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
435 440 445
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
450 455 460
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
465 470 475 480
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
485 490 495
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
500 505 510
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
515 520 525
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
530 535 540
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
545 550 555 560
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
565 570 575
Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
580 585 590
Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
595 600 605
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
610 615 620
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
625 630 635 640
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
645 650 655
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
660 665 670
<210> 456
<211> 447
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD20 VH-CH1(EE)-Fc (впадина, P329G LALA)
<400> 456
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Ala Phe Ser Tyr Ser
20 25 30
Trp Ile Asn Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Arg Ile Phe Pro Gly Asp Gly Asp Thr Asp Tyr Asn Gly Lys Phe
50 55 60
Lys Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asn Val Phe Asp Gly Tyr Trp Leu Val Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe
115 120 125
Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu
130 135 140
Gly Cys Leu Val Glu Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
145 150 155 160
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
165 170 175
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
180 185 190
Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro
195 200 205
Ser Asn Thr Lys Val Asp Glu Lys Val Glu Pro Lys Ser Cys Asp Lys
210 215 220
Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro
225 230 235 240
Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser
245 250 255
Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp
260 265 270
Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn
275 280 285
Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val
290 295 300
Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu
305 310 315 320
Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys
325 330 335
Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Cys Thr
340 345 350
Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Ser
355 360 365
Cys Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu
370 375 380
Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu
385 390 395 400
Asp Ser Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val Asp Lys
405 410 415
Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu
420 425 430
Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
<210> 457
<211> 219
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD20 VL-CL(RK)
<400> 457
Asp Ile Val Met Thr Gln Thr Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Lys Ser Leu Leu His Ser
20 25 30
Asn Gly Ile Thr Tyr Leu Tyr Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Gln Met Ser Asn Leu Val Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Ala Gln Asn
85 90 95
Leu Glu Leu Pro Tyr Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105 110
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Arg
115 120 125
Lys Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
130 135 140
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
145 150 155 160
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
165 170 175
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
180 185 190
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
195 200 205
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 458
<211> 232
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD3 VH-CL
<400> 458
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Thr Tyr
20 25 30
Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Arg Ile Arg Ser Lys Tyr Asn Asn Tyr Ala Thr Tyr Tyr Ala Asp
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Val Arg His Gly Asn Phe Gly Asn Ser Tyr Val Ser Trp Phe
100 105 110
Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Val
115 120 125
Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys
130 135 140
Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg
145 150 155 160
Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn
165 170 175
Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser
180 185 190
Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys
195 200 205
Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr
210 215 220
Lys Ser Phe Asn Arg Gly Glu Cys
225 230
<210> 459
<211> 290
<212> Белок
<213> Homo sapiens
<400> 459
Met Arg Ile Phe Ala Val Phe Ile Phe Met Thr Tyr Trp His Leu Leu
1 5 10 15
Asn Ala Phe Thr Val Thr Val Pro Lys Asp Leu Tyr Val Val Glu Tyr
20 25 30
Gly Ser Asn Met Thr Ile Glu Cys Lys Phe Pro Val Glu Lys Gln Leu
35 40 45
Asp Leu Ala Ala Leu Ile Val Tyr Trp Glu Met Glu Asp Lys Asn Ile
50 55 60
Ile Gln Phe Val His Gly Glu Glu Asp Leu Lys Val Gln His Ser Ser
65 70 75 80
Tyr Arg Gln Arg Ala Arg Leu Leu Lys Asp Gln Leu Ser Leu Gly Asn
85 90 95
Ala Ala Leu Gln Ile Thr Asp Val Lys Leu Gln Asp Ala Gly Val Tyr
100 105 110
Arg Cys Met Ile Ser Tyr Gly Gly Ala Asp Tyr Lys Arg Ile Thr Val
115 120 125
Lys Val Asn Ala Pro Tyr Asn Lys Ile Asn Gln Arg Ile Leu Val Val
130 135 140
Asp Pro Val Thr Ser Glu His Glu Leu Thr Cys Gln Ala Glu Gly Tyr
145 150 155 160
Pro Lys Ala Glu Val Ile Trp Thr Ser Ser Asp His Gln Val Leu Ser
165 170 175
Gly Lys Thr Thr Thr Thr Asn Ser Lys Arg Glu Glu Lys Leu Phe Asn
180 185 190
Val Thr Ser Thr Leu Arg Ile Asn Thr Thr Thr Asn Glu Ile Phe Tyr
195 200 205
Cys Thr Phe Arg Arg Leu Asp Pro Glu Glu Asn His Thr Ala Glu Leu
210 215 220
Val Ile Pro Glu Leu Pro Leu Ala His Pro Pro Asn Glu Arg Thr His
225 230 235 240
Leu Val Ile Leu Gly Ala Ile Leu Leu Cys Leu Gly Val Ala Leu Thr
245 250 255
Phe Ile Phe Arg Leu Arg Lys Gly Arg Met Met Asp Val Lys Lys Cys
260 265 270
Gly Ile Gln Asp Thr Asn Ser Lys Lys Gln Ser Asp Thr His Leu Glu
275 280 285
Glu Thr
290
<210> 460
<211> 118
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH (PD-L1)
<400> 460
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Ser
20 25 30
Trp Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Trp Ile Ser Pro Tyr Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Ala Asp Thr Ser Lys Asn Thr Ala Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Arg His Trp Pro Gly Gly Phe Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 461
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL (PD-L1)
<400> 461
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Asp Val Ser Thr Ala
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ser Ala Ser Phe Leu Tyr Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Leu Tyr His Pro Ala
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 462
<211> 121
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH (PD-L1) 2
<400> 462
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Arg Tyr
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Asn Ile Lys Gln Asp Gly Ser Glu Lys Tyr Tyr Val Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Gly Gly Trp Phe Gly Glu Leu Ala Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 463
<211> 108
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL (PD-L1) 2
<400> 463
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Arg Val Ser Ser Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Asp Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Ser Leu Pro
85 90 95
Trp Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 464
<211> 288
<212> Белок
<213> Homo sapiens
<400> 464
Met Gln Ile Pro Gln Ala Pro Trp Pro Val Val Trp Ala Val Leu Gln
1 5 10 15
Leu Gly Trp Arg Pro Gly Trp Phe Leu Asp Ser Pro Asp Arg Pro Trp
20 25 30
Asn Pro Pro Thr Phe Ser Pro Ala Leu Leu Val Val Thr Glu Gly Asp
35 40 45
Asn Ala Thr Phe Thr Cys Ser Phe Ser Asn Thr Ser Glu Ser Phe Val
50 55 60
Leu Asn Trp Tyr Arg Met Ser Pro Ser Asn Gln Thr Asp Lys Leu Ala
65 70 75 80
Ala Phe Pro Glu Asp Arg Ser Gln Pro Gly Gln Asp Cys Arg Phe Arg
85 90 95
Val Thr Gln Leu Pro Asn Gly Arg Asp Phe His Met Ser Val Val Arg
100 105 110
Ala Arg Arg Asn Asp Ser Gly Thr Tyr Leu Cys Gly Ala Ile Ser Leu
115 120 125
Ala Pro Lys Ala Gln Ile Lys Glu Ser Leu Arg Ala Glu Leu Arg Val
130 135 140
Thr Glu Arg Arg Ala Glu Val Pro Thr Ala His Pro Ser Pro Ser Pro
145 150 155 160
Arg Pro Ala Gly Gln Phe Gln Thr Leu Val Val Gly Val Val Gly Gly
165 170 175
Leu Leu Gly Ser Leu Val Leu Leu Val Trp Val Leu Ala Val Ile Cys
180 185 190
Ser Arg Ala Ala Arg Gly Thr Ile Gly Ala Arg Arg Thr Gly Gln Pro
195 200 205
Leu Lys Glu Asp Pro Ser Ala Val Pro Val Phe Ser Val Asp Tyr Gly
210 215 220
Glu Leu Asp Phe Gln Trp Arg Glu Lys Thr Pro Glu Pro Pro Val Pro
225 230 235 240
Cys Val Pro Glu Gln Thr Glu Tyr Ala Thr Ile Val Phe Pro Ser Gly
245 250 255
Met Gly Thr Ser Ser Pro Ala Arg Arg Gly Ser Ala Asp Gly Pro Arg
260 265 270
Ser Ala Gln Pro Leu Arg Pro Glu Asp Gly His Cys Ser Trp Pro Leu
275 280 285
<210> 465
<211> 120
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH (PD-1)
<400> 465
Gln Val Gln Leu Val Gln Ser Gly Val Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asn Tyr
20 25 30
Tyr Met Tyr Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Asn Pro Ser Asn Gly Gly Thr Asn Phe Asn Glu Lys Phe
50 55 60
Lys Asn Arg Val Thr Leu Thr Thr Asp Ser Ser Thr Thr Thr Ala Tyr
65 70 75 80
Met Glu Leu Lys Ser Leu Gln Phe Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Arg Asp Tyr Arg Phe Asp Met Gly Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 466
<211> 111
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL (PD-1)
<400> 466
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Lys Gly Val Ser Thr Ser
20 25 30
Gly Tyr Ser Tyr Leu His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro
35 40 45
Arg Leu Leu Ile Tyr Leu Ala Ser Tyr Leu Glu Ser Gly Val Pro Ala
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
65 70 75 80
Ser Leu Glu Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln His Ser Arg
85 90 95
Asp Leu Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105 110
<210> 467
<211> 113
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH (PD-1) 2
<400> 467
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Asp Cys Lys Ala Ser Gly Ile Thr Phe Ser Asn Ser
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Trp Tyr Asp Gly Ser Lys Arg Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Phe
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Asn Asp Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser
100 105 110
Ser
<210> 468
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL (PD-1) 2
<400> 468
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Arg Ala Thr Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Ser Ser Asn Trp Pro Arg
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 469
<211> 314
<212> Белок
<213> Homo sapiens
<400> 469
Met Ala Pro Pro Gln Val Leu Ala Phe Gly Leu Leu Leu Ala Ala Ala
1 5 10 15
Thr Ala Thr Phe Ala Ala Ala Gln Glu Glu Cys Val Cys Glu Asn Tyr
20 25 30
Lys Leu Ala Val Asn Cys Phe Val Asn Asn Asn Arg Gln Cys Gln Cys
35 40 45
Thr Ser Val Gly Ala Gln Asn Thr Val Ile Cys Ser Lys Leu Ala Ala
50 55 60
Lys Cys Leu Val Met Lys Ala Glu Met Asn Gly Ser Lys Leu Gly Arg
65 70 75 80
Arg Ala Lys Pro Glu Gly Ala Leu Gln Asn Asn Asp Gly Leu Tyr Asp
85 90 95
Pro Asp Cys Asp Glu Ser Gly Leu Phe Lys Ala Lys Gln Cys Asn Gly
100 105 110
Thr Ser Met Cys Trp Cys Val Asn Thr Ala Gly Val Arg Arg Thr Asp
115 120 125
Lys Asp Thr Glu Ile Thr Cys Ser Glu Arg Val Arg Thr Tyr Trp Ile
130 135 140
Ile Ile Glu Leu Lys His Lys Ala Arg Glu Lys Pro Tyr Asp Ser Lys
145 150 155 160
Ser Leu Arg Thr Ala Leu Gln Lys Glu Ile Thr Thr Arg Tyr Gln Leu
165 170 175
Asp Pro Lys Phe Ile Thr Ser Ile Leu Tyr Glu Asn Asn Val Ile Thr
180 185 190
Ile Asp Leu Val Gln Asn Ser Ser Gln Lys Thr Gln Asn Asp Val Asp
195 200 205
Ile Ala Asp Val Ala Tyr Tyr Phe Glu Lys Asp Val Lys Gly Glu Ser
210 215 220
Leu Phe His Ser Lys Lys Met Asp Leu Thr Val Asn Gly Glu Gln Leu
225 230 235 240
Asp Leu Asp Pro Gly Gln Thr Leu Ile Tyr Tyr Val Asp Glu Lys Ala
245 250 255
Pro Glu Phe Ser Met Gln Gly Leu Lys Ala Gly Val Ile Ala Val Ile
260 265 270
Val Val Val Val Ile Ala Val Val Ala Gly Ile Val Val Leu Val Ile
275 280 285
Ser Arg Lys Lys Arg Met Ala Lys Tyr Glu Lys Ala Glu Ile Lys Glu
290 295 300
Met Gly Glu Met His Arg Glu Leu Asn Ala
305 310
<210> 470
<211> 315
<212> Белок
<213> Mus musculus
<400> 470
Met Ala Gly Pro Gln Ala Leu Ala Phe Gly Leu Leu Leu Ala Val Val
1 5 10 15
Thr Ala Thr Leu Ala Ala Ala Gln Arg Asp Cys Val Cys Asp Asn Tyr
20 25 30
Lys Leu Ala Thr Ser Cys Ser Leu Asn Glu Tyr Gly Glu Cys Gln Cys
35 40 45
Thr Ser Tyr Gly Thr Gln Asn Thr Val Ile Cys Ser Lys Leu Ala Ser
50 55 60
Lys Cys Leu Ala Met Lys Ala Glu Met Thr His Ser Lys Ser Gly Arg
65 70 75 80
Arg Ile Lys Pro Glu Gly Ala Ile Gln Asn Asn Asp Gly Leu Tyr Asp
85 90 95
Pro Asp Cys Asp Glu Gln Gly Leu Phe Lys Ala Lys Gln Cys Asn Gly
100 105 110
Thr Ala Thr Cys Trp Cys Val Asn Thr Ala Gly Val Arg Arg Thr Asp
115 120 125
Lys Asp Thr Glu Ile Thr Cys Ser Glu Arg Val Arg Thr Tyr Trp Ile
130 135 140
Ile Ile Glu Leu Lys His Lys Glu Arg Glu Ser Pro Tyr Asp His Gln
145 150 155 160
Ser Leu Gln Thr Ala Leu Gln Glu Ala Phe Thr Ser Arg Tyr Lys Leu
165 170 175
Asn Gln Lys Phe Ile Lys Asn Ile Met Tyr Glu Asn Asn Val Ile Thr
180 185 190
Ile Asp Leu Met Gln Asn Ser Ser Gln Lys Thr Gln Asp Asp Val Asp
195 200 205
Ile Ala Asp Val Ala Tyr Tyr Phe Glu Lys Asp Val Lys Gly Glu Ser
210 215 220
Leu Phe His Ser Ser Lys Ser Met Asp Leu Arg Val Asn Gly Glu Pro
225 230 235 240
Leu Asp Leu Asp Pro Gly Gln Thr Leu Ile Tyr Tyr Val Asp Glu Lys
245 250 255
Ala Pro Glu Phe Ser Met Gln Gly Leu Thr Ala Gly Ile Ile Ala Val
260 265 270
Ile Val Val Val Ser Leu Ala Val Ile Ala Gly Ile Val Val Leu Val
275 280 285
Ile Ser Thr Arg Lys Lys Ser Ala Lys Tyr Glu Lys Ala Glu Ile Lys
290 295 300
Glu Met Gly Glu Ile His Arg Glu Leu Asn Ala
305 310 315
<210> 471
<211> 1342
<212> Белок
<213> Homo sapiens
<400> 471
Met Arg Ala Asn Asp Ala Leu Gln Val Leu Gly Leu Leu Phe Ser Leu
1 5 10 15
Ala Arg Gly Ser Glu Val Gly Asn Ser Gln Ala Val Cys Pro Gly Thr
20 25 30
Leu Asn Gly Leu Ser Val Thr Gly Asp Ala Glu Asn Gln Tyr Gln Thr
35 40 45
Leu Tyr Lys Leu Tyr Glu Arg Cys Glu Val Val Met Gly Asn Leu Glu
50 55 60
Ile Val Leu Thr Gly His Asn Ala Asp Leu Ser Phe Leu Gln Trp Ile
65 70 75 80
Arg Glu Val Thr Gly Tyr Val Leu Val Ala Met Asn Glu Phe Ser Thr
85 90 95
Leu Pro Leu Pro Asn Leu Arg Val Val Arg Gly Thr Gln Val Tyr Asp
100 105 110
Gly Lys Phe Ala Ile Phe Val Met Leu Asn Tyr Asn Thr Asn Ser Ser
115 120 125
His Ala Leu Arg Gln Leu Arg Leu Thr Gln Leu Thr Glu Ile Leu Ser
130 135 140
Gly Gly Val Tyr Ile Glu Lys Asn Asp Lys Leu Cys His Met Asp Thr
145 150 155 160
Ile Asp Trp Arg Asp Ile Val Arg Asp Arg Asp Ala Glu Ile Val Val
165 170 175
Lys Asp Asn Gly Arg Ser Cys Pro Pro Cys His Glu Val Cys Lys Gly
180 185 190
Arg Cys Trp Gly Pro Gly Ser Glu Asp Cys Gln Thr Leu Thr Lys Thr
195 200 205
Ile Cys Ala Pro Gln Cys Asn Gly His Cys Phe Gly Pro Asn Pro Asn
210 215 220
Gln Cys Cys His Asp Glu Cys Ala Gly Gly Cys Ser Gly Pro Gln Asp
225 230 235 240
Thr Asp Cys Phe Ala Cys Arg His Phe Asn Asp Ser Gly Ala Cys Val
245 250 255
Pro Arg Cys Pro Gln Pro Leu Val Tyr Asn Lys Leu Thr Phe Gln Leu
260 265 270
Glu Pro Asn Pro His Thr Lys Tyr Gln Tyr Gly Gly Val Cys Val Ala
275 280 285
Ser Cys Pro His Asn Phe Val Val Asp Gln Thr Ser Cys Val Arg Ala
290 295 300
Cys Pro Pro Asp Lys Met Glu Val Asp Lys Asn Gly Leu Lys Met Cys
305 310 315 320
Glu Pro Cys Gly Gly Leu Cys Pro Lys Ala Cys Glu Gly Thr Gly Ser
325 330 335
Gly Ser Arg Phe Gln Thr Val Asp Ser Ser Asn Ile Asp Gly Phe Val
340 345 350
Asn Cys Thr Lys Ile Leu Gly Asn Leu Asp Phe Leu Ile Thr Gly Leu
355 360 365
Asn Gly Asp Pro Trp His Lys Ile Pro Ala Leu Asp Pro Glu Lys Leu
370 375 380
Asn Val Phe Arg Thr Val Arg Glu Ile Thr Gly Tyr Leu Asn Ile Gln
385 390 395 400
Ser Trp Pro Pro His Met His Asn Phe Ser Val Phe Ser Asn Leu Thr
405 410 415
Thr Ile Gly Gly Arg Ser Leu Tyr Asn Arg Gly Phe Ser Leu Leu Ile
420 425 430
Met Lys Asn Leu Asn Val Thr Ser Leu Gly Phe Arg Ser Leu Lys Glu
435 440 445
Ile Ser Ala Gly Arg Ile Tyr Ile Ser Ala Asn Arg Gln Leu Cys Tyr
450 455 460
His His Ser Leu Asn Trp Thr Lys Val Leu Arg Gly Pro Thr Glu Glu
465 470 475 480
Arg Leu Asp Ile Lys His Asn Arg Pro Arg Arg Asp Cys Val Ala Glu
485 490 495
Gly Lys Val Cys Asp Pro Leu Cys Ser Ser Gly Gly Cys Trp Gly Pro
500 505 510
Gly Pro Gly Gln Cys Leu Ser Cys Arg Asn Tyr Ser Arg Gly Gly Val
515 520 525
Cys Val Thr His Cys Asn Phe Leu Asn Gly Glu Pro Arg Glu Phe Ala
530 535 540
His Glu Ala Glu Cys Phe Ser Cys His Pro Glu Cys Gln Pro Met Glu
545 550 555 560
Gly Thr Ala Thr Cys Asn Gly Ser Gly Ser Asp Thr Cys Ala Gln Cys
565 570 575
Ala His Phe Arg Asp Gly Pro His Cys Val Ser Ser Cys Pro His Gly
580 585 590
Val Leu Gly Ala Lys Gly Pro Ile Tyr Lys Tyr Pro Asp Val Gln Asn
595 600 605
Glu Cys Arg Pro Cys His Glu Asn Cys Thr Gln Gly Cys Lys Gly Pro
610 615 620
Glu Leu Gln Asp Cys Leu Gly Gln Thr Leu Val Leu Ile Gly Lys Thr
625 630 635 640
His Leu Thr Met Ala Leu Thr Val Ile Ala Gly Leu Val Val Ile Phe
645 650 655
Met Met Leu Gly Gly Thr Phe Leu Tyr Trp Arg Gly Arg Arg Ile Gln
660 665 670
Asn Lys Arg Ala Met Arg Arg Tyr Leu Glu Arg Gly Glu Ser Ile Glu
675 680 685
Pro Leu Asp Pro Ser Glu Lys Ala Asn Lys Val Leu Ala Arg Ile Phe
690 695 700
Lys Glu Thr Glu Leu Arg Lys Leu Lys Val Leu Gly Ser Gly Val Phe
705 710 715 720
Gly Thr Val His Lys Gly Val Trp Ile Pro Glu Gly Glu Ser Ile Lys
725 730 735
Ile Pro Val Cys Ile Lys Val Ile Glu Asp Lys Ser Gly Arg Gln Ser
740 745 750
Phe Gln Ala Val Thr Asp His Met Leu Ala Ile Gly Ser Leu Asp His
755 760 765
Ala His Ile Val Arg Leu Leu Gly Leu Cys Pro Gly Ser Ser Leu Gln
770 775 780
Leu Val Thr Gln Tyr Leu Pro Leu Gly Ser Leu Leu Asp His Val Arg
785 790 795 800
Gln His Arg Gly Ala Leu Gly Pro Gln Leu Leu Leu Asn Trp Gly Val
805 810 815
Gln Ile Ala Lys Gly Met Tyr Tyr Leu Glu Glu His Gly Met Val His
820 825 830
Arg Asn Leu Ala Ala Arg Asn Val Leu Leu Lys Ser Pro Ser Gln Val
835 840 845
Gln Val Ala Asp Phe Gly Val Ala Asp Leu Leu Pro Pro Asp Asp Lys
850 855 860
Gln Leu Leu Tyr Ser Glu Ala Lys Thr Pro Ile Lys Trp Met Ala Leu
865 870 875 880
Glu Ser Ile His Phe Gly Lys Tyr Thr His Gln Ser Asp Val Trp Ser
885 890 895
Tyr Gly Val Thr Val Trp Glu Leu Met Thr Phe Gly Ala Glu Pro Tyr
900 905 910
Ala Gly Leu Arg Leu Ala Glu Val Pro Asp Leu Leu Glu Lys Gly Glu
915 920 925
Arg Leu Ala Gln Pro Gln Ile Cys Thr Ile Asp Val Tyr Met Val Met
930 935 940
Val Lys Cys Trp Met Ile Asp Glu Asn Ile Arg Pro Thr Phe Lys Glu
945 950 955 960
Leu Ala Asn Glu Phe Thr Arg Met Ala Arg Asp Pro Pro Arg Tyr Leu
965 970 975
Val Ile Lys Arg Glu Ser Gly Pro Gly Ile Ala Pro Gly Pro Glu Pro
980 985 990
His Gly Leu Thr Asn Lys Lys Leu Glu Glu Val Glu Leu Glu Pro Glu
995 1000 1005
Leu Asp Leu Asp Leu Asp Leu Glu Ala Glu Glu Asp Asn Leu Ala
1010 1015 1020
Thr Thr Thr Leu Gly Ser Ala Leu Ser Leu Pro Val Gly Thr Leu
1025 1030 1035
Asn Arg Pro Arg Gly Ser Gln Ser Leu Leu Ser Pro Ser Ser Gly
1040 1045 1050
Tyr Met Pro Met Asn Gln Gly Asn Leu Gly Glu Ser Cys Gln Glu
1055 1060 1065
Ser Ala Val Ser Gly Ser Ser Glu Arg Cys Pro Arg Pro Val Ser
1070 1075 1080
Leu His Pro Met Pro Arg Gly Cys Leu Ala Ser Glu Ser Ser Glu
1085 1090 1095
Gly His Val Thr Gly Ser Glu Ala Glu Leu Gln Glu Lys Val Ser
1100 1105 1110
Met Cys Arg Ser Arg Ser Arg Ser Arg Ser Pro Arg Pro Arg Gly
1115 1120 1125
Asp Ser Ala Tyr His Ser Gln Arg His Ser Leu Leu Thr Pro Val
1130 1135 1140
Thr Pro Leu Ser Pro Pro Gly Leu Glu Glu Glu Asp Val Asn Gly
1145 1150 1155
Tyr Val Met Pro Asp Thr His Leu Lys Gly Thr Pro Ser Ser Arg
1160 1165 1170
Glu Gly Thr Leu Ser Ser Val Gly Leu Ser Ser Val Leu Gly Thr
1175 1180 1185
Glu Glu Glu Asp Glu Asp Glu Glu Tyr Glu Tyr Met Asn Arg Arg
1190 1195 1200
Arg Arg His Ser Pro Pro His Pro Pro Arg Pro Ser Ser Leu Glu
1205 1210 1215
Glu Leu Gly Tyr Glu Tyr Met Asp Val Gly Ser Asp Leu Ser Ala
1220 1225 1230
Ser Leu Gly Ser Thr Gln Ser Cys Pro Leu His Pro Val Pro Ile
1235 1240 1245
Met Pro Thr Ala Gly Thr Thr Pro Asp Glu Asp Tyr Glu Tyr Met
1250 1255 1260
Asn Arg Gln Arg Asp Gly Gly Gly Pro Gly Gly Asp Tyr Ala Ala
1265 1270 1275
Met Gly Ala Cys Pro Ala Ser Glu Gln Gly Tyr Glu Glu Met Arg
1280 1285 1290
Ala Phe Gln Gly Pro Gly His Gln Ala Pro His Val His Tyr Ala
1295 1300 1305
Arg Leu Lys Thr Leu Arg Ser Leu Glu Ala Thr Asp Ser Ala Phe
1310 1315 1320
Asp Asn Pro Asp Tyr Trp His Ser Arg Leu Phe Pro Lys Ala Asn
1325 1330 1335
Ala Gln Arg Thr
1340
<210> 472
<211> 595
<212> Белок
<213> Homo sapiens
<400> 472
Met Arg Val Leu Leu Ala Ala Leu Gly Leu Leu Phe Leu Gly Ala Leu
1 5 10 15
Arg Ala Phe Pro Gln Asp Arg Pro Phe Glu Asp Thr Cys His Gly Asn
20 25 30
Pro Ser His Tyr Tyr Asp Lys Ala Val Arg Arg Cys Cys Tyr Arg Cys
35 40 45
Pro Met Gly Leu Phe Pro Thr Gln Gln Cys Pro Gln Arg Pro Thr Asp
50 55 60
Cys Arg Lys Gln Cys Glu Pro Asp Tyr Tyr Leu Asp Glu Ala Asp Arg
65 70 75 80
Cys Thr Ala Cys Val Thr Cys Ser Arg Asp Asp Leu Val Glu Lys Thr
85 90 95
Pro Cys Ala Trp Asn Ser Ser Arg Val Cys Glu Cys Arg Pro Gly Met
100 105 110
Phe Cys Ser Thr Ser Ala Val Asn Ser Cys Ala Arg Cys Phe Phe His
115 120 125
Ser Val Cys Pro Ala Gly Met Ile Val Lys Phe Pro Gly Thr Ala Gln
130 135 140
Lys Asn Thr Val Cys Glu Pro Ala Ser Pro Gly Val Ser Pro Ala Cys
145 150 155 160
Ala Ser Pro Glu Asn Cys Lys Glu Pro Ser Ser Gly Thr Ile Pro Gln
165 170 175
Ala Lys Pro Thr Pro Val Ser Pro Ala Thr Ser Ser Ala Ser Thr Met
180 185 190
Pro Val Arg Gly Gly Thr Arg Leu Ala Gln Glu Ala Ala Ser Lys Leu
195 200 205
Thr Arg Ala Pro Asp Ser Pro Ser Ser Val Gly Arg Pro Ser Ser Asp
210 215 220
Pro Gly Leu Ser Pro Thr Gln Pro Cys Pro Glu Gly Ser Gly Asp Cys
225 230 235 240
Arg Lys Gln Cys Glu Pro Asp Tyr Tyr Leu Asp Glu Ala Gly Arg Cys
245 250 255
Thr Ala Cys Val Ser Cys Ser Arg Asp Asp Leu Val Glu Lys Thr Pro
260 265 270
Cys Ala Trp Asn Ser Ser Arg Thr Cys Glu Cys Arg Pro Gly Met Ile
275 280 285
Cys Ala Thr Ser Ala Thr Asn Ser Cys Ala Arg Cys Val Pro Tyr Pro
290 295 300
Ile Cys Ala Ala Glu Thr Val Thr Lys Pro Gln Asp Met Ala Glu Lys
305 310 315 320
Asp Thr Thr Phe Glu Ala Pro Pro Leu Gly Thr Gln Pro Asp Cys Asn
325 330 335
Pro Thr Pro Glu Asn Gly Glu Ala Pro Ala Ser Thr Ser Pro Thr Gln
340 345 350
Ser Leu Leu Val Asp Ser Gln Ala Ser Lys Thr Leu Pro Ile Pro Thr
355 360 365
Ser Ala Pro Val Ala Leu Ser Ser Thr Gly Lys Pro Val Leu Asp Ala
370 375 380
Gly Pro Val Leu Phe Trp Val Ile Leu Val Leu Val Val Val Val Gly
385 390 395 400
Ser Ser Ala Phe Leu Leu Cys His Arg Arg Ala Cys Arg Lys Arg Ile
405 410 415
Arg Gln Lys Leu His Leu Cys Tyr Pro Val Gln Thr Ser Gln Pro Lys
420 425 430
Leu Glu Leu Val Asp Ser Arg Pro Arg Arg Ser Ser Thr Gln Leu Arg
435 440 445
Ser Gly Ala Ser Val Thr Glu Pro Val Ala Glu Glu Arg Gly Leu Met
450 455 460
Ser Gln Pro Leu Met Glu Thr Cys His Ser Val Gly Ala Ala Tyr Leu
465 470 475 480
Glu Ser Leu Pro Leu Gln Asp Ala Ser Pro Ala Gly Gly Pro Ser Ser
485 490 495
Pro Arg Asp Leu Pro Glu Pro Arg Val Ser Thr Glu His Thr Asn Asn
500 505 510
Lys Ile Glu Lys Ile Tyr Ile Met Lys Ala Asp Thr Val Ile Val Gly
515 520 525
Thr Val Lys Ala Glu Leu Pro Glu Gly Arg Gly Leu Ala Gly Pro Ala
530 535 540
Glu Pro Glu Leu Glu Glu Glu Leu Glu Ala Asp His Thr Pro His Tyr
545 550 555 560
Pro Glu Gln Glu Thr Glu Pro Pro Leu Gly Ser Cys Ser Asp Val Met
565 570 575
Leu Ser Val Glu Glu Glu Gly Lys Glu Asp Pro Leu Pro Thr Ala Ala
580 585 590
Ser Gly Lys
595
<210> 473
<211> 420
<212> Белок
<213> Homo sapiens
<400> 473
Met Pro Gly Gly Cys Ser Arg Gly Pro Ala Ala Gly Asp Gly Arg Leu
1 5 10 15
Arg Leu Ala Arg Leu Ala Leu Val Leu Leu Gly Trp Val Ser Ser Ser
20 25 30
Ser Pro Thr Ser Ser Ala Ser Ser Phe Ser Ser Ser Ala Pro Phe Leu
35 40 45
Ala Ser Ala Val Ser Ala Gln Pro Pro Leu Pro Asp Gln Cys Pro Ala
50 55 60
Leu Cys Glu Cys Ser Glu Ala Ala Arg Thr Val Lys Cys Val Asn Arg
65 70 75 80
Asn Leu Thr Glu Val Pro Thr Asp Leu Pro Ala Tyr Val Arg Asn Leu
85 90 95
Phe Leu Thr Gly Asn Gln Leu Ala Val Leu Pro Ala Gly Ala Phe Ala
100 105 110
Arg Arg Pro Pro Leu Ala Glu Leu Ala Ala Leu Asn Leu Ser Gly Ser
115 120 125
Arg Leu Asp Glu Val Arg Ala Gly Ala Phe Glu His Leu Pro Ser Leu
130 135 140
Arg Gln Leu Asp Leu Ser His Asn Pro Leu Ala Asp Leu Ser Pro Phe
145 150 155 160
Ala Phe Ser Gly Ser Asn Ala Ser Val Ser Ala Pro Ser Pro Leu Val
165 170 175
Glu Leu Ile Leu Asn His Ile Val Pro Pro Glu Asp Glu Arg Gln Asn
180 185 190
Arg Ser Phe Glu Gly Met Val Val Ala Ala Leu Leu Ala Gly Arg Ala
195 200 205
Leu Gln Gly Leu Arg Arg Leu Glu Leu Ala Ser Asn His Phe Leu Tyr
210 215 220
Leu Pro Arg Asp Val Leu Ala Gln Leu Pro Ser Leu Arg His Leu Asp
225 230 235 240
Leu Ser Asn Asn Ser Leu Val Ser Leu Thr Tyr Val Ser Phe Arg Asn
245 250 255
Leu Thr His Leu Glu Ser Leu His Leu Glu Asp Asn Ala Leu Lys Val
260 265 270
Leu His Asn Gly Thr Leu Ala Glu Leu Gln Gly Leu Pro His Ile Arg
275 280 285
Val Phe Leu Asp Asn Asn Pro Trp Val Cys Asp Cys His Met Ala Asp
290 295 300
Met Val Thr Trp Leu Lys Glu Thr Glu Val Val Gln Gly Lys Asp Arg
305 310 315 320
Leu Thr Cys Ala Tyr Pro Glu Lys Met Arg Asn Arg Val Leu Leu Glu
325 330 335
Leu Asn Ser Ala Asp Leu Asp Cys Asp Pro Ile Leu Pro Pro Ser Leu
340 345 350
Gln Thr Ser Tyr Val Phe Leu Gly Ile Val Leu Ala Leu Ile Gly Ala
355 360 365
Ile Phe Leu Leu Val Leu Tyr Leu Asn Arg Lys Gly Ile Lys Lys Trp
370 375 380
Met His Asn Ile Arg Asp Ala Cys Arg Asp His Met Glu Gly Tyr His
385 390 395 400
Tyr Arg Tyr Glu Ile Asn Ala Asp Pro Arg Leu Thr Asn Leu Ser Ser
405 410 415
Asn Ser Asp Val
420
<210> 474
<211> 300
<212> Белок
<213> Homo sapiens
<400> 474
Met Ala Asn Cys Glu Phe Ser Pro Val Ser Gly Asp Lys Pro Cys Cys
1 5 10 15
Arg Leu Ser Arg Arg Ala Gln Leu Cys Leu Gly Val Ser Ile Leu Val
20 25 30
Leu Ile Leu Val Val Val Leu Ala Val Val Val Pro Arg Trp Arg Gln
35 40 45
Gln Trp Ser Gly Pro Gly Thr Thr Lys Arg Phe Pro Glu Thr Val Leu
50 55 60
Ala Arg Cys Val Lys Tyr Thr Glu Ile His Pro Glu Met Arg His Val
65 70 75 80
Asp Cys Gln Ser Val Trp Asp Ala Phe Lys Gly Ala Phe Ile Ser Lys
85 90 95
His Pro Cys Asn Ile Thr Glu Glu Asp Tyr Gln Pro Leu Met Lys Leu
100 105 110
Gly Thr Gln Thr Val Pro Cys Asn Lys Ile Leu Leu Trp Ser Arg Ile
115 120 125
Lys Asp Leu Ala His Gln Phe Thr Gln Val Gln Arg Asp Met Phe Thr
130 135 140
Leu Glu Asp Thr Leu Leu Gly Tyr Leu Ala Asp Asp Leu Thr Trp Cys
145 150 155 160
Gly Glu Phe Asn Thr Ser Lys Ile Asn Tyr Gln Ser Cys Pro Asp Trp
165 170 175
Arg Lys Asp Cys Ser Asn Asn Pro Val Ser Val Phe Trp Lys Thr Val
180 185 190
Ser Arg Arg Phe Ala Glu Ala Ala Cys Asp Val Val His Val Met Leu
195 200 205
Asn Gly Ser Arg Ser Lys Ile Phe Asp Lys Asn Ser Thr Phe Gly Ser
210 215 220
Val Glu Val His Asn Leu Gln Pro Glu Lys Val Gln Thr Leu Glu Ala
225 230 235 240
Trp Val Ile His Gly Gly Arg Glu Asp Ser Arg Asp Leu Cys Gln Asp
245 250 255
Pro Thr Ile Lys Glu Leu Glu Ser Ile Ile Ser Lys Arg Asn Ile Gln
260 265 270
Phe Ser Cys Lys Asn Ile Tyr Arg Pro Asp Lys Phe Leu Gln Cys Val
275 280 285
Lys Asn Pro Glu Asp Ser Ser Cys Thr Ser Glu Ile
290 295 300
<210> 475
<211> 184
<212> Белок
<213> Homo sapiens
<400> 475
Met Leu Gln Met Ala Gly Gln Cys Ser Gln Asn Glu Tyr Phe Asp Ser
1 5 10 15
Leu Leu His Ala Cys Ile Pro Cys Gln Leu Arg Cys Ser Ser Asn Thr
20 25 30
Pro Pro Leu Thr Cys Gln Arg Tyr Cys Asn Ala Ser Val Thr Asn Ser
35 40 45
Val Lys Gly Thr Asn Ala Ile Leu Trp Thr Cys Leu Gly Leu Ser Leu
50 55 60
Ile Ile Ser Leu Ala Val Phe Val Leu Met Phe Leu Leu Arg Lys Ile
65 70 75 80
Asn Ser Glu Pro Leu Lys Asp Glu Phe Lys Asn Thr Gly Ser Gly Leu
85 90 95
Leu Gly Met Ala Asn Ile Asp Leu Glu Lys Ser Arg Thr Gly Asp Glu
100 105 110
Ile Ile Leu Pro Arg Gly Leu Glu Tyr Thr Val Glu Glu Cys Thr Cys
115 120 125
Glu Asp Cys Ile Lys Ser Lys Pro Lys Val Asp Ser Asp His Cys Phe
130 135 140
Pro Leu Pro Ala Met Glu Glu Gly Ala Thr Ile Leu Val Thr Thr Lys
145 150 155 160
Thr Asn Asp Tyr Cys Lys Ser Leu Pro Ala Ala Leu Ser Ala Thr Glu
165 170 175
Ile Glu Lys Ser Ile Ser Ala Arg
180
<210> 476
<211> 345
<212> Белок
<213> Homo sapiens
<400> 476
Met Tyr Lys Asp Cys Ile Glu Ser Thr Gly Asp Tyr Phe Leu Leu Cys
1 5 10 15
Asp Ala Glu Gly Pro Trp Gly Ile Ile Leu Glu Ser Leu Ala Ile Leu
20 25 30
Gly Ile Val Val Thr Ile Leu Leu Leu Leu Ala Phe Leu Phe Leu Met
35 40 45
Arg Lys Ile Gln Asp Cys Ser Gln Trp Asn Val Leu Pro Thr Gln Leu
50 55 60
Leu Phe Leu Leu Ser Val Leu Gly Leu Phe Gly Leu Ala Phe Ala Phe
65 70 75 80
Ile Ile Glu Leu Asn Gln Gln Thr Ala Pro Val Arg Tyr Phe Leu Phe
85 90 95
Gly Val Leu Phe Ala Leu Cys Phe Ser Cys Leu Leu Ala His Ala Ser
100 105 110
Asn Leu Val Lys Leu Val Arg Gly Cys Val Ser Phe Ser Trp Thr Thr
115 120 125
Ile Leu Cys Ile Ala Ile Gly Cys Ser Leu Leu Gln Ile Ile Ile Ala
130 135 140
Thr Glu Tyr Val Thr Leu Ile Met Thr Arg Gly Met Met Phe Val Asn
145 150 155 160
Met Thr Pro Cys Gln Leu Asn Val Asp Phe Val Val Leu Leu Val Tyr
165 170 175
Val Leu Phe Leu Met Ala Leu Thr Phe Phe Val Ser Lys Ala Thr Phe
180 185 190
Cys Gly Pro Cys Glu Asn Trp Lys Gln His Gly Arg Leu Ile Phe Ile
195 200 205
Thr Val Leu Phe Ser Ile Ile Ile Trp Val Val Trp Ile Ser Met Leu
210 215 220
Leu Arg Gly Asn Pro Gln Phe Gln Arg Gln Pro Gln Trp Asp Asp Pro
225 230 235 240
Val Val Cys Ile Ala Leu Val Thr Asn Ala Trp Val Phe Leu Leu Leu
245 250 255
Tyr Ile Val Pro Glu Leu Cys Ile Leu Tyr Arg Ser Cys Arg Gln Glu
260 265 270
Cys Pro Leu Gln Gly Asn Ala Cys Pro Val Thr Ala Tyr Gln His Ser
275 280 285
Phe Gln Val Glu Asn Gln Glu Leu Ser Arg Ala Arg Asp Ser Asp Gly
290 295 300
Ala Glu Glu Asp Val Ala Leu Thr Ser Tyr Gly Thr Pro Ile Gln Pro
305 310 315 320
Gln Thr Val Asp Pro Thr Gln Glu Cys Phe Ile Pro Gln Ala Lys Leu
325 330 335
Ser Pro Gln Gln Asp Ala Gly Gly Val
340 345
<210> 477
<211> 98
<212> Белок
<213> Homo sapiens
<400> 477
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys
1 5 10 15
Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr
65 70 75 80
Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Lys Val
<210> 478
<211> 107
<212> Белок
<213> Homo sapiens
<400> 478
Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
1 5 10 15
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
20 25 30
Trp Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val
35 40 45
Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln
50 55 60
Glu Ser Thr Tyr Arg Trp Ser Val Leu Thr Val Leu His Gln Asp Trp
65 70 75 80
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
85 90 95
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys
100 105
<210> 479
<211> 106
<212> Белок
<213> Homo sapiens
<400> 479
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp
1 5 10 15
Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
20 25 30
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
35 40 45
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
50 55 60
Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly
65 70 75 80
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
85 90 95
Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly
100 105
<210> 480
<211> 5
<212> Белок
<213> Homo sapiens
<400> 480
Glu Pro Lys Ser Cys
1 5
<210> 481
<211> 10
<212> Белок
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<222> (8)..(8)
<223> X is S или P
<400> 481
Asp Lys Thr His Thr Cys Pro Xaa Cys Pro
1 5 10
<210> 482
<211> 7
<212> Белок
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<222> (5)..(5)
<223> X is S или P
<400> 482
His Thr Cys Pro Xaa Cys Pro
1 5
<210> 483
<211> 5
<212> Белок
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<222> (3)..(3)
<223> X is S или P
<400> 483
Cys Pro Xaa Cys Pro
1 5
<210> 484
<211> 330
<212> Белок
<213> Homo sapiens
<400> 484
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys
1 5 10 15
Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr
65 70 75 80
Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys
100 105 110
Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro
115 120 125
Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys
130 135 140
Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp
145 150 155 160
Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu
165 170 175
Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu
180 185 190
His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn
195 200 205
Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly
210 215 220
Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu
225 230 235 240
Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr
245 250 255
Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn
260 265 270
Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe
275 280 285
Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn
290 295 300
Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr
305 310 315 320
Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
325 330
<210> 485
<211> 330
<212> Белок
<213> Homo sapiens
<400> 485
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys
1 5 10 15
Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr
65 70 75 80
Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys
100 105 110
Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro
115 120 125
Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys
130 135 140
Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp
145 150 155 160
Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu
165 170 175
Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu
180 185 190
His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn
195 200 205
Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly
210 215 220
Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu
225 230 235 240
Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr
245 250 255
Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn
260 265 270
Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe
275 280 285
Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn
290 295 300
Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr
305 310 315 320
Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
325 330
<210> 486
<211> 326
<212> Белок
<213> Homo sapiens
<400> 486
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg
1 5 10 15
Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Asn Phe Gly Thr Gln Thr
65 70 75 80
Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Thr Val Glu Arg Lys Cys Cys Val Glu Cys Pro Pro Cys Pro Ala Pro
100 105 110
Pro Val Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
115 120 125
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
130 135 140
Val Ser His Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly
145 150 155 160
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn
165 170 175
Ser Thr Phe Arg Val Val Ser Val Leu Thr Val Val His Gln Asp Trp
180 185 190
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro
195 200 205
Ala Pro Ile Glu Lys Thr Ile Ser Lys Thr Lys Gly Gln Pro Arg Glu
210 215 220
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn
225 230 235 240
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
245 250 255
Ser Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
260 265 270
Thr Pro Pro Met Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
275 280 285
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
290 295 300
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
305 310 315 320
Ser Leu Ser Pro Gly Lys
325
<210> 487
<211> 377
<212> Белок
<213> Homo sapiens
<400> 487
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg
1 5 10 15
Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr
65 70 75 80
Tyr Thr Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Arg Val Glu Leu Lys Thr Pro Leu Gly Asp Thr Thr His Thr Cys Pro
100 105 110
Arg Cys Pro Glu Pro Lys Ser Cys Asp Thr Pro Pro Pro Cys Pro Arg
115 120 125
Cys Pro Glu Pro Lys Ser Cys Asp Thr Pro Pro Pro Cys Pro Arg Cys
130 135 140
Pro Glu Pro Lys Ser Cys Asp Thr Pro Pro Pro Cys Pro Arg Cys Pro
145 150 155 160
Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
165 170 175
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
180 185 190
Val Val Asp Val Ser His Glu Asp Pro Glu Val Gln Phe Lys Trp Tyr
195 200 205
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
210 215 220
Gln Tyr Asn Ser Thr Phe Arg Val Val Ser Val Leu Thr Val Leu His
225 230 235 240
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
245 250 255
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Thr Lys Gly Gln
260 265 270
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met
275 280 285
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
290 295 300
Ser Asp Ile Ala Val Glu Trp Glu Ser Ser Gly Gln Pro Glu Asn Asn
305 310 315 320
Tyr Asn Thr Thr Pro Pro Met Leu Asp Ser Asp Gly Ser Phe Phe Leu
325 330 335
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Ile
340 345 350
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn Arg Phe Thr Gln
355 360 365
Lys Ser Leu Ser Leu Ser Pro Gly Lys
370 375
<210> 488
<211> 327
<212> Белок
<213> Homo sapiens
<400> 488
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg
1 5 10 15
Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Lys Thr
65 70 75 80
Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Arg Val Glu Ser Lys Tyr Gly Pro Pro Cys Pro Ser Cys Pro Ala Pro
100 105 110
Glu Phe Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
115 120 125
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
130 135 140
Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp
145 150 155 160
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe
165 170 175
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
180 185 190
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu
195 200 205
Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
210 215 220
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys
225 230 235 240
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
245 250 255
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
260 265 270
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
275 280 285
Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser
290 295 300
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
305 310 315 320
Leu Ser Leu Ser Leu Gly Lys
325
<210> 489
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(вариант 8) CDR-H1
<400> 489
Ser Tyr Tyr Ile His
1 5
<210> 490
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(вариант 8) CDR-H2
<400> 490
Ser Ile Tyr Pro Gly Asn Val Gln Thr Asn Tyr Asn Glu Lys Phe Lys
1 5 10 15
Asp
<210> 491
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(вариант 8) CDR-H3
<400> 491
Ser His Tyr Gly Leu Asp Trp Asn Phe Asp Val
1 5 10
<210> 492
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(вариант 8) CDR-L1
<400> 492
His Ala Ser Gln Asn Ile Tyr Val Tyr Leu Asn
1 5 10
<210> 493
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(вариант 8) CDR-L2
<400> 493
Lys Ala Ser Asn Leu His Thr
1 5
<210> 494
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(вариант 8) CDR-L3
<400> 494
Gln Gln Gly Gln Thr Tyr Pro Tyr Thr
1 5
<210> 495
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(вариант 15) CDR-H1
<400> 495
Ser Tyr Tyr Ile His
1 5
<210> 496
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(вариант 15) CDR-H2
<400> 496
Ser Ile Tyr Pro Gly Asn Val Gln Thr Asn Tyr Asn Glu Lys Phe Lys
1 5 10 15
Asp
<210> 497
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(вариант 15) CDR-H3
<400> 497
Ser His Tyr Gly Leu Asp Trp Asn Phe Asp Val
1 5 10
<210> 498
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(вариант 15) CDR-L1
<400> 498
His Ala Ser Gln Asn Ile Tyr Val Phe Leu Asn
1 5 10
<210> 499
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(вариант 15) CDR-L2
<400> 499
Lys Ala Ser Asn Leu His Thr
1 5
<210> 500
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(вариант 15) CDR-L3
<400> 500
Gln Gln Gly Gln Thr Tyr Pro Tyr Thr
1 5
<210> 501
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(вариант 29) CDR-H1
<400> 501
Ser Tyr Tyr Ile His
1 5
<210> 502
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(вариант 29) CDR-H2
<400> 502
Ser Ile Tyr Pro Gly Asn Val Asn Thr Asn Tyr Asn Glu Lys Phe Lys
1 5 10 15
Asp
<210> 503
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(вариант 29) CDR-H3
<400> 503
Ser His Tyr Gly Leu Asp Trp Asn Phe Asp Val
1 5 10
<210> 504
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(вариант 29) CDR-L1
<400> 504
His Ala Ser Gln Asn Ile Tyr Val Trp Leu Asn
1 5 10
<210> 505
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(вариант 29) CDR-L2
<400> 505
Lys Ala Ser Asn Leu His Thr
1 5
<210> 506
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD28(вариант 29) CDR-L3
<400> 506
Gln Gln Gly Gln Thr Tyr Pro Tyr Thr
1 5
<210> 507
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (T84.66-LCHA)- CDR-H1
<400> 507
Asp Thr Tyr Met His
1 5
<210> 508
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (T84.66-LCHA)- CDR-H2
<400> 508
Arg Ile Asp Pro Ala Asn Gly Asn Ser Lys Tyr Val Pro Lys Phe Gln
1 5 10 15
Gly
<210> 509
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (T84.66-LCHA)- CDR-H3
<400> 509
Phe Gly Tyr Tyr Val Ser Asp Tyr Ala Met Ala Tyr
1 5 10
<210> 510
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (T84.66-LCHA)- CDR-L1
<400> 510
Arg Ala Gly Glu Ser Val Asp Ile Phe Gly Val Gly Phe Leu His
1 5 10 15
<210> 511
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (T84.66-LCHA)- CDR-L2
<400> 511
Arg Ala Ser Asn Arg Ala Thr
1 5
<210> 512
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (T84.66-LCHA)- CDR-L3
<400> 512
Gln Gln Thr Asn Glu Asp Pro Tyr Thr
1 5
<210> 513
<211> 121
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (T84.66-LCHA) VH
<400> 513
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Phe Asn Ile Lys Asp Thr
20 25 30
Tyr Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Arg Ile Asp Pro Ala Asn Gly Asn Ser Lys Tyr Val Pro Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Pro Phe Gly Tyr Tyr Val Ser Asp Tyr Ala Met Ala Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 514
<211> 111
<212> Белок
<213> Искусственная последовательность
<220>
<223> CEA (T84.66-LCHA) VL
<400> 514
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Gly Glu Ser Val Asp Ile Phe
20 25 30
Gly Val Gly Phe Leu His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro
35 40 45
Arg Leu Leu Ile Tyr Arg Ala Ser Asn Arg Ala Thr Gly Ile Pro Ala
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
65 70 75 80
Ser Leu Glu Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Thr Asn
85 90 95
Glu Asp Pro Tyr Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105 110
<210> 515
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> EpCAM (MT201)- CDR-H1
<400> 515
Ser Tyr Gly Met His
1 5
<210> 516
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> EpCAM (MT201)- CDR-H2
<400> 516
Val Ile Ser Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 517
<211> 16
<212> Белок
<213> Искусственная последовательность
<220>
<223> EpCAM (MT201)- CDR-H3
<400> 517
Asp Met Gly Trp Gly Ser Gly Trp Arg Pro Tyr Tyr Tyr Tyr Gly Met
1 5 10 15
<210> 518
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> EpCAM (MT201)- CDR-L1
<400> 518
Arg Thr Ser Gln Ser Ile Ser Ser Tyr Leu Asn
1 5 10
<210> 519
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> EpCAM (MT201)- CDR-L2
<400> 519
Trp Ala Ser Thr Arg Glu Ser
1 5
<210> 520
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> EpCAM (MT201)- CDR-L3
<400> 520
Gln Gln Ser Tyr Asp Ile Pro Tyr Thr
1 5
<210> 521
<211> 127
<212> Белок
<213> Искусственная последовательность
<220>
<223> EpCAM (MT201) VH
<400> 521
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Ser Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Asp Met Gly Trp Gly Ser Gly Trp Arg Pro Tyr Tyr Tyr Tyr
100 105 110
Gly Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120 125
<210> 522
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> EpCAM (MT201) VL
<400> 522
Glu Leu Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Thr Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro Lys Leu Leu Ile
35 40 45
Tyr Trp Ala Ser Thr Arg Glu Ser Gly Val Pro Asp Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ser Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Asp Ile Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 523
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> HER3- CDR-H1
<400> 523
Ser Ser Tyr Ile Ser
1 5
<210> 524
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> HER3- CDR-H2
<400> 524
Trp Ile Tyr Ala Gly Thr Gly Ser Pro Ser Tyr Asn Gln Lys Leu Gln
1 5 10 15
Gly
<210> 525
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> HER3- CDR-H3
<400> 525
His Arg Asp Tyr Tyr Ser Asn Ser Leu
1 5
<210> 526
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> HER3- CDR-L1
<400> 526
Lys Ser Ser Gln Ser Val Leu Asn Ser Gly Asn Gln Lys Asn Tyr Leu
1 5 10 15
Thr
<210> 527
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> HER3- CDR-L2
<400> 527
Trp Ala Ser Thr Arg Glu Ser
1 5
<210> 528
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> HER3- CDR-L3
<400> 528
Gln Ser Asp Tyr Ser Tyr Pro Tyr Thr
1 5
<210> 529
<211> 120
<212> Белок
<213> Искусственная последовательность
<220>
<223> HER3 VH
<400> 529
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Arg Ser Ser
20 25 30
Tyr Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Tyr Ala Gly Thr Gly Ser Pro Ser Tyr Asn Gln Lys Leu
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg His Arg Asp Tyr Tyr Ser Asn Ser Leu Thr Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 530
<211> 113
<212> Белок
<213> Искусственная последовательность
<220>
<223> HER3 VL
<400> 530
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Ile Asn Cys Lys Ser Ser Gln Ser Val Leu Asn Ser
20 25 30
Gly Asn Gln Lys Asn Tyr Leu Thr Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Pro Pro Lys Leu Leu Ile Tyr Trp Ala Ser Thr Arg Glu Ser Gly Val
50 55 60
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
65 70 75 80
Ile Ser Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Tyr Cys Gln Ser
85 90 95
Asp Tyr Ser Tyr Pro Tyr Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile
100 105 110
Lys
<210> 531
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD30- CDR-H1
<400> 531
Asp Tyr Tyr Ile Thr
1 5
<210> 532
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD30- CDR-H2
<400> 532
Trp Ile Tyr Pro Gly Ser Gly Asn Thr Lys Tyr Asn Glu Lys Phe Lys
1 5 10 15
Gly
<210> 533
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD30- CDR-H3
<400> 533
Tyr Gly Asn Tyr Trp Phe
1 5
<210> 534
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD30- CDR-L1
<400> 534
Lys Ala Ser Gln Ser Val Asp Phe Asp Gly Asp Ser Tyr Met Asn
1 5 10 15
<210> 535
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD30- CDR-L2
<400> 535
Ala Ala Ser Thr Leu Gln Ile
1 5
<210> 536
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD30- CDR-L3
<400> 536
Gln Gln Ser Asn Glu Asp Pro Trp Thr
1 5
<210> 537
<211> 117
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD30 VH
<400> 537
Gln Ile Gln Leu Gln Gln Ser Gly Pro Glu Val Val Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Ile Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Tyr Ile Thr Trp Val Lys Gln Lys Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Trp Ile Tyr Pro Gly Ser Gly Asn Thr Lys Tyr Asn Glu Lys Phe
50 55 60
Lys Gly Lys Ala Thr Leu Thr Val Asp Thr Ser Ser Ser Thr Ala Phe
65 70 75 80
Met Gln Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val Tyr Phe Cys
85 90 95
Ala Asn Tyr Gly Asn Tyr Trp Phe Ala Tyr Trp Gly Gln Gly Thr Gln
100 105 110
Val Thr Val Ser Ala
115
<210> 538
<211> 111
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD30 VL
<400> 538
Asp Ile Val Leu Thr Gln Ser Pro Ala Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Gln Arg Ala Thr Ile Ser Cys Lys Ala Ser Gln Ser Val Asp Phe Asp
20 25 30
Gly Asp Ser Tyr Met Asn Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro
35 40 45
Lys Val Leu Ile Tyr Ala Ala Ser Asn Leu Glu Ser Gly Ile Pro Ala
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Asn Ile His
65 70 75 80
Pro Val Glu Glu Glu Asp Ala Ala Thr Tyr Tyr Cys Gln Gln Ser Asn
85 90 95
Glu Asp Pro Trp Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105 110
<210> 539
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> TPBG(FAB091)- CDR-H1
<400> 539
Ser Asp Ala Met His
1 5
<210> 540
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> TPBG(FAB091)- CDR-H2
<400> 540
Gly Val Ser Gly Ser Gly Gly Ser Pro Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 541
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> TPBG(FAB091)- CDR-H3
<400> 541
Gly Gly Ser Ile Ala Gly Ser Tyr Tyr Tyr Tyr Pro Met Asp Val
1 5 10 15
<210> 542
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> TPBG(FAB091)- CDR-L1
<400> 542
Gln Ala Ser Gln Asp Ile Ser Asn Tyr Leu Asn
1 5 10
<210> 543
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> TPBG(FAB091)- CDR-L2
<400> 543
Ala Ala Ser Thr Leu Gln Ile
1 5
<210> 544
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> TPBG(FAB091)- CDR-L3
<400> 544
Gln Gln Ala Asn Ser Phe Pro Leu Thr
1 5
<210> 545
<211> 124
<212> Белок
<213> Искусственная последовательность
<220>
<223> TPBG(FAB091) VH
<400> 545
Glu Val His Leu Leu Glu Ser Gly Gly Gly Leu Val His Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Arg Ser Asp
20 25 30
Ala Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Val Ser Gly Ser Gly Gly Ser Pro Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Thr Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Gly Gly Ser Ile Ala Gly Ser Tyr Tyr Tyr Tyr Pro Met Asp
100 105 110
Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 546
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> TPBG(FAB091) VL
<400> 546
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asp Ile Ser Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Gln Ile Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ala Asn Ser Phe Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 547
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD38- CDR-H1
<400> 547
Ser Phe Ala Met Ser
1 5
<210> 548
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD38- CDR-H2
<400> 548
Ala Ile Ser Gly Ser Gly Gly Gly Thr Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 549
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD38- CDR-H3
<400> 549
Asp Lys Ile Leu Trp Phe Gly Glu Pro Val Phe Asp Tyr
1 5 10
<210> 550
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD38- CDR-L1
<400> 550
Arg Ala Ser Gln Ser Val Ser Ser Tyr Leu Ala
1 5 10
<210> 551
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD38- CDR-L2
<400> 551
Asp Ala Ser Asn Arg Ala Thr
1 5
<210> 552
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD38- CDR-L3
<400> 552
Gln Gln Arg Ser Asn Trp Pro Pro Thr
1 5
<210> 553
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD38 VH
<400> 553
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Val Ser Gly Phe Thr Phe Asn Ser Phe
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Gly Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Phe Cys
85 90 95
Ala Lys Asp Lys Ile Leu Trp Phe Gly Glu Pro Val Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 554
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD38 VL
<400> 554
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Arg Ala Thr Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Arg Ser Asn Trp Pro Pro
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 555
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> BCMA - CDR-H1
<400> 555
Ser Tyr Ala Met Asn
1 5
<210> 556
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> BCMA - CDR-H2
<400> 556
Ala Ile Thr Ala Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 557
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> BCMA - CDR-H3
<400> 557
Tyr Trp Pro Met Ser Leu
1 5
<210> 558
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> BCMA - CDR-L1
<400> 558
Arg Ala Ser Gln Ser Val Ser Ala Tyr Tyr Leu Ala
1 5 10
<210> 559
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> BCMA - CDR-L2
<400> 559
Asp Ala Ser Ile Arg Ala Thr
1 5
<210> 560
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> BCMA - CDR-L3
<400> 560
Gln Gln Tyr Glu Arg Trp Pro Leu Thr
1 5
<210> 561
<211> 115
<212> Белок
<213> Искусственная последовательность
<220>
<223> BCMA VH
<400> 561
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Thr Ala Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Tyr Trp Pro Met Ser Leu Trp Gly Gln Gly Thr Leu Val Thr
100 105 110
Val Ser Ser
115
<210> 562
<211> 108
<212> Белок
<213> Искусственная последовательность
<220>
<223> BCMA VL
<400> 562
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ala Tyr
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Met Tyr Asp Ala Ser Ile Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Glu Arg Trp Pro
85 90 95
Leu Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 563
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> GPRC5D (5E11) - CDR-H1
<400> 563
Lys Tyr Ala Met Ala
1 5
<210> 564
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> GPRC5D (5E11) - CDR-H2
<400> 564
Ser Ile Ser Thr Gly Gly Val Asn Thr Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 565
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> GPRC5D (5E11) - CDR-H3
<400> 565
His Thr Gly Asp Tyr Phe Asp Tyr
1 5
<210> 566
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> GPRC5D (5E11) - CDR-L1
<400> 566
Arg Ala Ser Gln Ser Val Ser Ile Ser Gly Ile Asn Leu Met Asn
1 5 10 15
<210> 567
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> GPRC5D (5E11) - CDR-L2
<400> 567
His Ala Ser Ile Leu Ala Ser
1 5
<210> 568
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> GPRC5D (5E11) - CDR-L3
<400> 568
Gln Gln Thr Arg Glu Ser Pro Leu Thr
1 5
<210> 569
<211> 117
<212> Белок
<213> Искусственная последовательность
<220>
<223> GPRC5D (5E11) VH1c
<400> 569
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Lys Tyr
20 25 30
Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Ser Ile Ser Thr Gly Gly Val Asn Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr His Thr Gly Asp Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Met
100 105 110
Val Thr Val Ser Ser
115
<210> 570
<211> 111
<212> Белок
<213> Искусственная последовательность
<220>
<223> GPRC5D (5E11) VL2b
<400> 570
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ile Ser
20 25 30
Gly Ile Asn Leu Met Asn Trp Tyr Gln Gln Lys Pro Gly Gln Gln Pro
35 40 45
Lys Leu Leu Ile Tyr His Ala Ser Ile Leu Ala Ser Gly Ile Pro Asp
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
65 70 75 80
Arg Leu Glu Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Thr Arg
85 90 95
Glu Ser Pro Leu Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys
100 105 110
<210> 571
<211> 117
<212> Белок
<213> Искусственная последовательность
<220>
<223> GPRC5D (5E11) VH1a
<400> 571
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Lys Tyr
20 25 30
Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Ser Ile Ser Thr Gly Gly Val Asn Thr Tyr Tyr Arg Asp Ser Val
50 55 60
Lys Ala Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr His Thr Gly Asp Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Met
100 105 110
Val Thr Val Ser Ser
115
<210> 572
<211> 117
<212> Белок
<213> Искусственная последовательность
<220>
<223> GPRC5D (5E11) VH1b
<400> 572
Glu Leu Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Lys Tyr
20 25 30
Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Ser Ile Ser Thr Gly Gly Val Asn Thr Tyr Tyr Arg Asp Ser Val
50 55 60
Lys Ala Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr His Thr Gly Asp Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Met
100 105 110
Val Thr Val Ser Ser
115
<210> 573
<211> 117
<212> Белок
<213> Искусственная последовательность
<220>
<223> GPRC5D (5E11) VH1d
<400> 573
Glu Leu Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Lys Tyr
20 25 30
Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Ser Ile Ser Thr Gly Gly Val Asn Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr His Thr Gly Asp Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Met
100 105 110
Val Thr Val Ser Ser
115
<210> 574
<211> 111
<212> Белок
<213> Искусственная последовательность
<220>
<223> GPRC5D (5E11) VL1a
<400> 574
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Ile Asn Cys Arg Ala Ser Gln Ser Val Ser Ile Ser
20 25 30
Gly Ile Asn Leu Met Asn Trp Tyr Gln Gln Lys Pro Gly Gln Gln Pro
35 40 45
Lys Leu Leu Ile Tyr His Ala Ser Ile Leu Ala Ser Gly Val Pro Asp
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
65 70 75 80
Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Tyr Cys Gln Gln Thr Arg
85 90 95
Glu Ser Pro Leu Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys
100 105 110
<210> 575
<211> 111
<212> Белок
<213> Искусственная последовательность
<220>
<223> GPRC5D (5E11) VL1c
<400> 575
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Ile Asn Cys Lys Ser Ser Gln Ser Val Ser Ile Ser
20 25 30
Gly Ile Asn Leu Met Asn Trp Tyr Gln Gln Lys Pro Gly Gln Gln Pro
35 40 45
Lys Leu Leu Ile Tyr His Ala Ser Ile Leu Ala Ser Gly Val Pro Asp
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
65 70 75 80
Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Tyr Cys Gln Gln Thr Arg
85 90 95
Glu Ser Pro Leu Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys
100 105 110
<210> 576
<211> 111
<212> Белок
<213> Искусственная последовательность
<220>
<223> GPRC5D (5E11) VL2a
<400> 576
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ile Ser
20 25 30
Gly Ile Asn Leu Met Asn Trp Tyr Gln Gln Lys Pro Gly Gln Gln Pro
35 40 45
Arg Leu Leu Ile Tyr His Ala Ser Ile Leu Ala Ser Gly Ile Pro Asp
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
65 70 75 80
Arg Leu Glu Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Thr Arg
85 90 95
Glu Ser Pro Leu Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys
100 105 110
<210> 577
<211> 111
<212> Белок
<213> Искусственная последовательность
<220>
<223> GPRC5D (5E11) VL3a
<400> 577
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Val Ser Ile Ser
20 25 30
Gly Ile Asn Leu Met Asn Trp Tyr Gln Gln Lys Pro Gly Lys Gln Pro
35 40 45
Lys Leu Leu Ile Tyr His Ala Ser Ile Leu Ala Ser Gly Val Pro Ser
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
65 70 75 80
Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Thr Arg
85 90 95
Glu Ser Pro Leu Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys
100 105 110
<210> 578
<211> 111
<212> Белок
<213> Искусственная последовательность
<220>
<223> GPRC5D (5E11) VL3b
<400> 578
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Val Ser Ile Ser
20 25 30
Gly Ile Asn Leu Met Asn Trp Tyr Gln Gln Lys Pro Gly Gln Gln Pro
35 40 45
Lys Leu Leu Ile Tyr His Ala Ser Ile Leu Ala Ser Gly Val Pro Ser
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
65 70 75 80
Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Thr Arg
85 90 95
Glu Ser Pro Leu Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys
100 105 110
<210> 579
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> GPRC5D (5F11) - CDR-H1
<400> 579
Asn Tyr Gly Met Ala
1 5
<210> 580
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> GPRC5D (5F11) - CDR-H2
<400> 580
Ser Ile Ser Thr Gly Gly Gly Asn Thr Tyr Tyr Arg Asp Ser Val Lys
1 5 10 15
Gly
<210> 581
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> GPRC5D (5F11) - CDR-H3
<400> 581
His Asp Arg Gly Gly Leu Tyr
1 5
<210> 582
<211> 16
<212> Белок
<213> Искусственная последовательность
<220>
<223> GPRC5D (5F11) - CDR-L1
<400> 582
Arg Ser Ser Lys Ser Leu Leu His Ser Asn Gly Ile Thr Tyr Val Tyr
1 5 10 15
<210> 583
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> GPRC5D (5F11) - CDR-L2
<400> 583
Arg Met Ser Asn Arg Ala Ser
1 5
<210> 584
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> GPRC5D (5F11) - CDR-L3
<400> 584
Gly Gln Leu Leu Glu Asn Pro Tyr Thr
1 5
<210> 585
<211> 116
<212> Белок
<213> Искусственная последовательность
<220>
<223> GPRC5D (5F11) VH1a
<400> 585
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser Phe Ser Asn Tyr
20 25 30
Gly Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Ser Ile Ser Thr Gly Gly Gly Asn Thr Tyr Tyr Arg Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Arg His Asp Arg Gly Gly Leu Tyr Trp Gly Gln Gly Thr Met Val
100 105 110
Thr Val Ser Ser
115
<210> 586
<211> 116
<212> Белок
<213> Искусственная последовательность
<220>
<223> GPRC5D (5F11) VH1b
<400> 586
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser Phe Ser Asn Tyr
20 25 30
Gly Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Ser Ile Ser Thr Gly Gly Gly Asn Thr Tyr Tyr Arg Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Arg His Asp Arg Gly Gly Leu Tyr Trp Gly Gln Gly Thr Met Val
100 105 110
Thr Val Ser Ser
115
<210> 587
<211> 116
<212> Белок
<213> Искусственная последовательность
<220>
<223> GPRC5D (5F11) VH1c
<400> 587
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser Phe Ser Asn Tyr
20 25 30
Gly Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Ser Ile Ser Thr Gly Gly Gly Asn Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Arg His Asp Arg Gly Gly Leu Tyr Trp Gly Gln Gly Thr Met Val
100 105 110
Thr Val Ser Ser
115
<210> 588
<211> 116
<212> Белок
<213> Искусственная последовательность
<220>
<223> GPRC5D (5F11) VH1d
<400> 588
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser Phe Ser Asn Tyr
20 25 30
Gly Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Ser Ile Ser Thr Gly Gly Gly Asn Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Arg His Asp Arg Gly Gly Leu Tyr Trp Gly Gln Gly Thr Met Val
100 105 110
Thr Val Ser Ser
115
<210> 589
<211> 116
<212> Белок
<213> Искусственная последовательность
<220>
<223> GPRC5D (5F11) VH2b
<400> 589
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser Phe Ser Asn Tyr
20 25 30
Gly Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Ser Ile Ser Thr Gly Gly Gly Asn Thr Tyr Tyr Arg Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Arg His Asp Arg Gly Gly Leu Tyr Trp Gly Gln Gly Thr Met Val
100 105 110
Thr Val Ser Ser
115
<210> 590
<211> 116
<212> Белок
<213> Искусственная последовательность
<220>
<223> GPRC5D (5F11) VH2d
<400> 590
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser Phe Ser Asn Tyr
20 25 30
Gly Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Ser Ile Ser Thr Gly Gly Gly Asn Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Arg His Asp Arg Gly Gly Leu Tyr Trp Gly Gln Gly Thr Met Val
100 105 110
Thr Val Ser Ser
115
<210> 591
<211> 112
<212> Белок
<213> Искусственная последовательность
<220>
<223> GPRC5D (5F11) VL1a
<400> 591
Asp Ile Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Lys Ser Leu Leu His Ser
20 25 30
Asn Gly Ile Thr Tyr Val Tyr Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Val Leu Ile Tyr Arg Met Ser Asn Leu Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr His Cys Gly Gln Leu
85 90 95
Leu Glu Asn Pro Tyr Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105 110
<210> 592
<211> 112
<212> Белок
<213> Искусственная последовательность
<220>
<223> GPRC5D (5F11) VL1b
<400> 592
Asp Ile Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Lys Ser Leu Leu His Ser
20 25 30
Asn Gly Ile Thr Tyr Val Tyr Trp Tyr Leu Gln Lys Pro Gly Lys Ser
35 40 45
Pro Gln Val Leu Ile Tyr Arg Met Ser Asn Leu Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr His Cys Gly Gln Leu
85 90 95
Leu Glu Asn Pro Tyr Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105 110
<210> 593
<211> 112
<212> Белок
<213> Искусственная последовательность
<220>
<223> GPRC5D (5F11) VL2a
<400> 593
Asp Ile Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Lys Ser Leu Leu His Ser
20 25 30
Asn Gly Ile Thr Tyr Val Tyr Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Arg Met Ser Asn Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr His Cys Gly Gln Leu
85 90 95
Leu Glu Asn Pro Tyr Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105 110
<210> 594
<211> 112
<212> Белок
<213> Искусственная последовательность
<220>
<223> GPRC5D (5F11) VL2b
<400> 594
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Ile Asn Cys Lys Ser Ser Lys Ser Leu Leu His Ser
20 25 30
Asn Gly Ile Thr Tyr Val Tyr Trp Tyr Gln Gln Lys Pro Gly Gln Pro
35 40 45
Pro Lys Leu Leu Ile Tyr Arg Met Ser Asn Leu Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile
65 70 75 80
Ser Ser Leu Gln Ala Glu Asp Val Ala Val Tyr His Cys Gly Gln Leu
85 90 95
Leu Glu Asn Pro Tyr Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105 110
<210> 595
<211> 112
<212> Белок
<213> Искусственная последовательность
<220>
<223> GPRC5D (5F11) VL2c
<400> 595
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Lys Ser Leu Leu His Ser
20 25 30
Asn Gly Ile Thr Tyr Val Tyr Trp Tyr Gln Gln Lys Pro Gly Gln Ala
35 40 45
Pro Arg Leu Leu Ile Tyr Arg Met Ser Asn Leu Ala Ser Gly Ile Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile
65 70 75 80
Ser Arg Leu Glu Pro Glu Asp Phe Ala Val Tyr His Cys Gly Gln Leu
85 90 95
Leu Glu Asn Pro Tyr Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105 110
<210> 596
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD3 (Cl22) CDR-H1
<400> 596
Ser Tyr Ala Met Asn
1 5
<210> 597
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD3 (Cl22) CDR-H2
<400> 597
Arg Ile Arg Ser Lys Tyr Asn Asn Tyr Ala Thr Tyr Tyr Ala Asp Ser
1 5 10 15
Val Lys Gly
<210> 598
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD3 (Cl22) CDR-H3
<400> 598
His Thr Thr Phe Pro Ser Ser Tyr Val Ser Tyr Tyr Gly Tyr
1 5 10
<210> 599
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD3 (Cl22) CDR-L1
<400> 599
Gly Ser Ser Thr Gly Ala Val Thr Thr Ser Asn Tyr Ala Asn
1 5 10
<210> 600
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD3 (Cl22) CDR-L2
<400> 600
Gly Thr Asn Lys Arg Ala Pro
1 5
<210> 601
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD3 (Cl22) CDR-L3
<400> 601
Ala Leu Trp Tyr Ser Asn Leu Trp Val
1 5
<210> 602
<211> 125
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD3 (Cl22) VH
<400> 602
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Gln Phe Ser Ser Tyr
20 25 30
Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Arg Ile Arg Ser Lys Tyr Asn Asn Tyr Ala Thr Tyr Tyr Ala Asp
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Val Arg His Thr Thr Phe Pro Ser Ser Tyr Val Ser Tyr Tyr
100 105 110
Gly Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 603
<211> 109
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD3 (Cl22) VL
<400> 603
Gln Ala Val Val Thr Gln Glu Pro Ser Leu Thr Val Ser Pro Gly Gly
1 5 10 15
Thr Val Thr Leu Thr Cys Gly Ser Ser Thr Gly Ala Val Thr Thr Ser
20 25 30
Asn Tyr Ala Asn Trp Val Gln Glu Lys Pro Gly Gln Ala Phe Arg Gly
35 40 45
Leu Ile Gly Gly Thr Asn Lys Arg Ala Pro Gly Thr Pro Ala Arg Phe
50 55 60
Ser Gly Ser Leu Leu Gly Gly Lys Ala Ala Leu Thr Leu Ser Gly Ala
65 70 75 80
Gln Pro Glu Asp Glu Ala Glu Tyr Tyr Cys Ala Leu Trp Tyr Ser Asn
85 90 95
Leu Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105
<210> 604
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD3 (V9) CDR-H1
<400> 604
Gly Tyr Ser Phe Thr Gly Tyr Thr Met Asn
1 5 10
<210> 605
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD3 (V9) CDR-H2
<400> 605
Leu Ile Asn Pro Tyr Lys Gly Val Ser Thr Tyr Asn Gln Lys Phe Lys
1 5 10 15
Asp
<210> 606
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD3 (V9) CDR-H3
<400> 606
Ser Gly Tyr Tyr Gly Asp Ser Asp Trp Tyr Phe Asp Val
1 5 10
<210> 607
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD3 (V9) CDR-L1
<400> 607
Arg Ala Ser Gln Asp Ile Arg Asn Tyr Leu Asn
1 5 10
<210> 608
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD3 (V9) CDR-L2
<400> 608
Tyr Thr Ser Arg Leu Glu Ser
1 5
<210> 609
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD3 (V9) CDR-L3
<400> 609
Gln Gln Gly Asn Thr Leu Pro Trp Thr
1 5
<210> 610
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD3 (V9) VH
<400> 610
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Tyr Ser Phe Thr Gly Tyr
20 25 30
Thr Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Leu Ile Asn Pro Tyr Lys Gly Val Ser Thr Tyr Asn Gln Lys Phe
50 55 60
Lys Asp Arg Phe Thr Ile Ser Val Asp Lys Ser Lys Asn Thr Ala Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Gly Tyr Tyr Gly Asp Ser Asp Trp Tyr Phe Asp Val Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 611
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD3 (V9) VL
<400> 611
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Asp Ile Arg Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Tyr Thr Ser Arg Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gly Asn Thr Leu Pro Trp
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| НАЦЕЛЕННЫЕ НА ОПУХОЛЬ АГОНИСТИЧЕСКИЕ CD28-АНТИГЕНСВЯЗЫВАЮЩИЕ МОЛЕКУЛЫ | 2019 |
|
RU2837821C1 |
| ТРИСПЕЦИФИЧЕСКИЕ СВЯЗЫВАЮЩИЕ БЕЛКИ, ОТНОСЯЩИЕСЯ К НИМ СПОСОБЫ И ВАРИАНТЫ ИХ ПРИМЕНЕНИЯ | 2020 |
|
RU2822200C2 |
| УЛУЧШЕННЫЕ АНТИГЕНСВЯЗЫВАЮЩИЕ РЕЦЕПТОРЫ | 2018 |
|
RU2825816C2 |
| ТЕРАПЕВТИЧЕСКИЕ АНТИТЕЛА ПРОТИВ ЛИГАНДА CD40 | 2018 |
|
RU2770209C2 |
| Выделенный альтернативный внутриклеточный сигнальный домен химерного антигенного рецептора и включающий его химерный антигенный рецептор | 2019 |
|
RU2742000C2 |
| ТРИСПЕЦИФИЧЕСКИЕ И/ИЛИ ТРИВАЛЕНТНЫЕ СВЯЗЫВАЮЩИЕ БЕЛКИ С ПРИМЕНЕНИЕМ КРОССОВЕРНОГО ФОРМАТА С ДВОЙНЫМ ВАРИАБЕЛЬНЫМ ДОМЕНОМ (CODV) ДЛЯ ЛЕЧЕНИЯ ИНФЕКЦИИ, ВЫЗВАННОЙ HIV | 2020 |
|
RU2820164C2 |
| АНТИТЕЛА К CD38 И КОМБИНАЦИИ С АНТИТЕЛАМИ К CD3 И CD28 | 2018 |
|
RU2812910C2 |
| КАНИНИЗИРОВАННЫЕ АНТИТЕЛА ПРОТИВ ЧЕЛОВЕЧЕСКОГО CTLA-4 | 2020 |
|
RU2822460C2 |
| АНТИТЕЛА ПРОТИВ СОБАЧЬЕГО CTLA-4 | 2020 |
|
RU2818586C2 |
| ТРИСПЕЦИФИЧЕСКИЕ СВЯЗЫВАЮЩИЕ БЕЛКИ, СВЯЗЫВАЮЩИЕ CD38, CD28 И CD3, А ТАКЖЕ СПОСОБЫ ПРИМЕНЕНИЯ ДЛЯ ЛЕЧЕНИЯ ВИРУСНОЙ ИНФЕКЦИИ | 2019 |
|
RU2820351C2 |
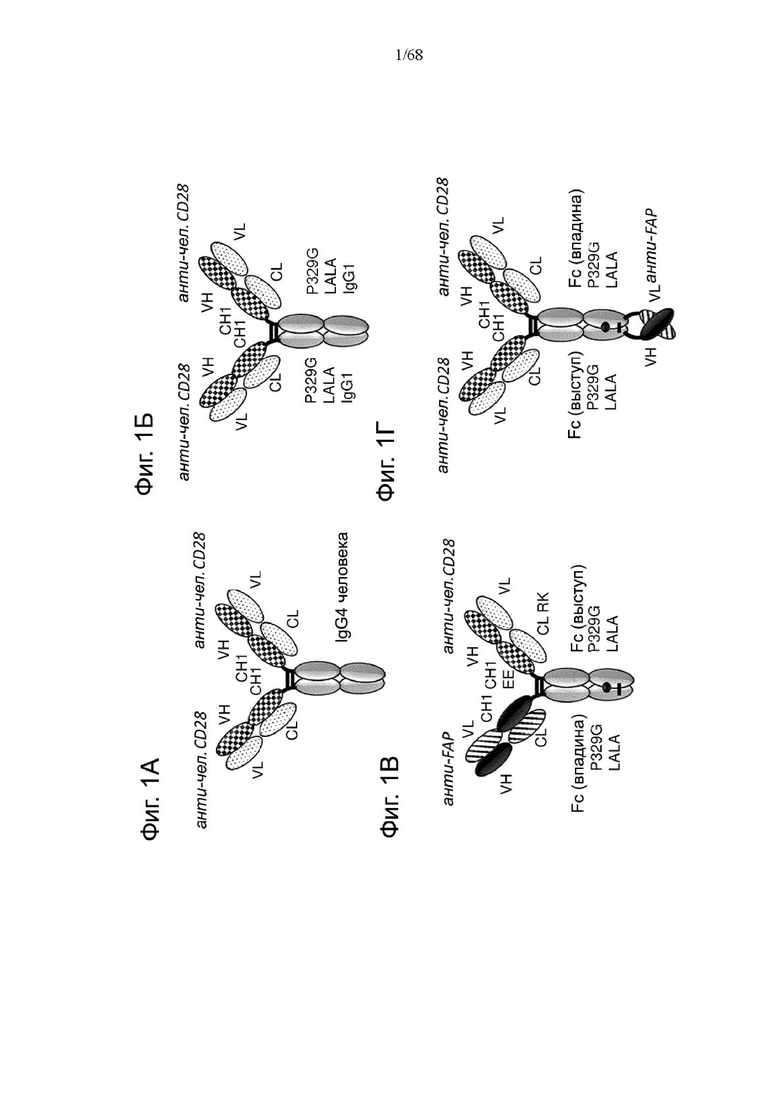
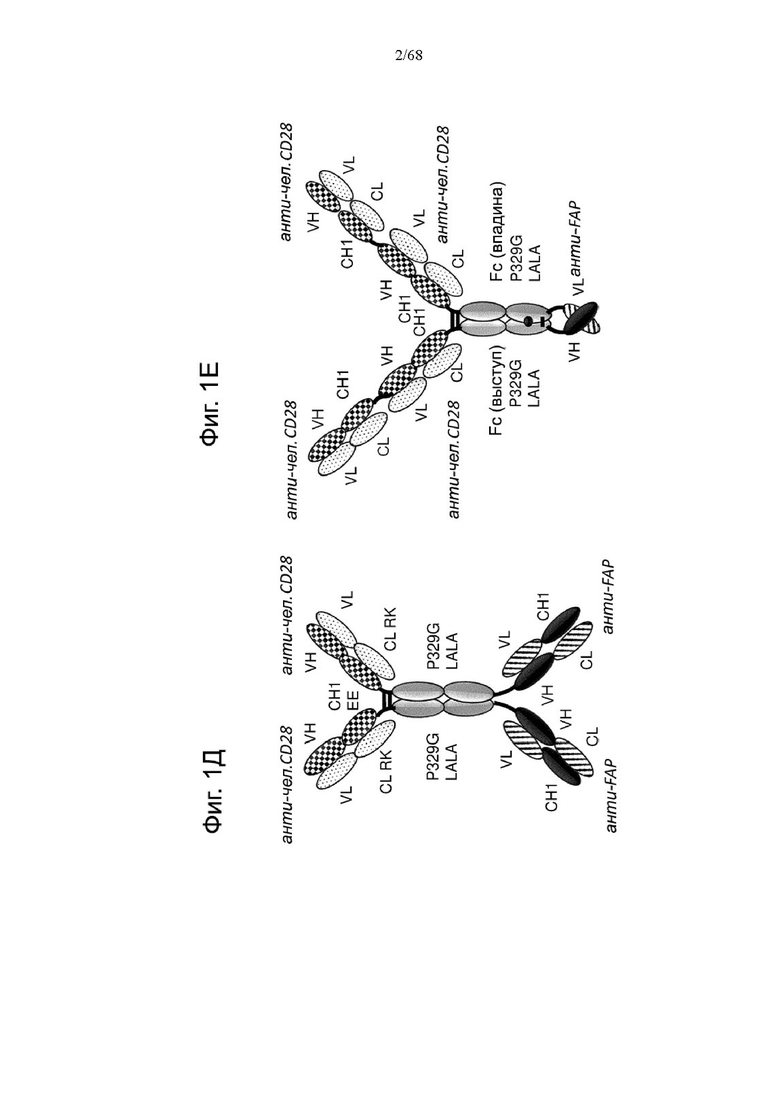
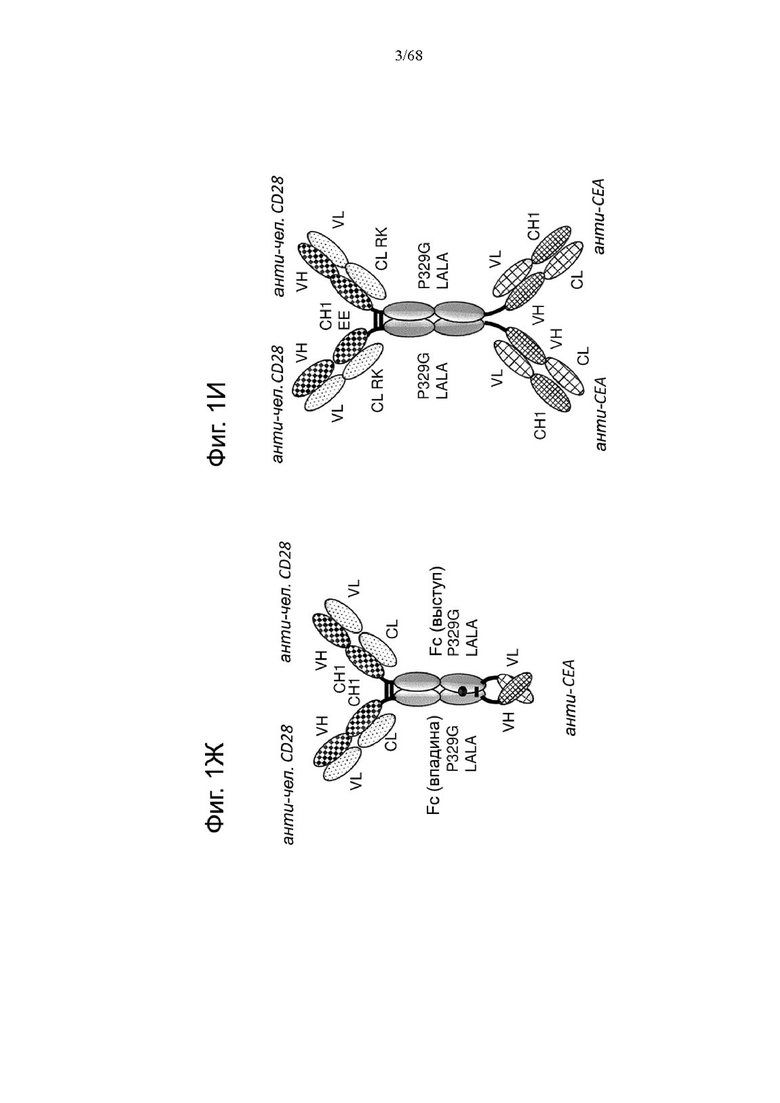
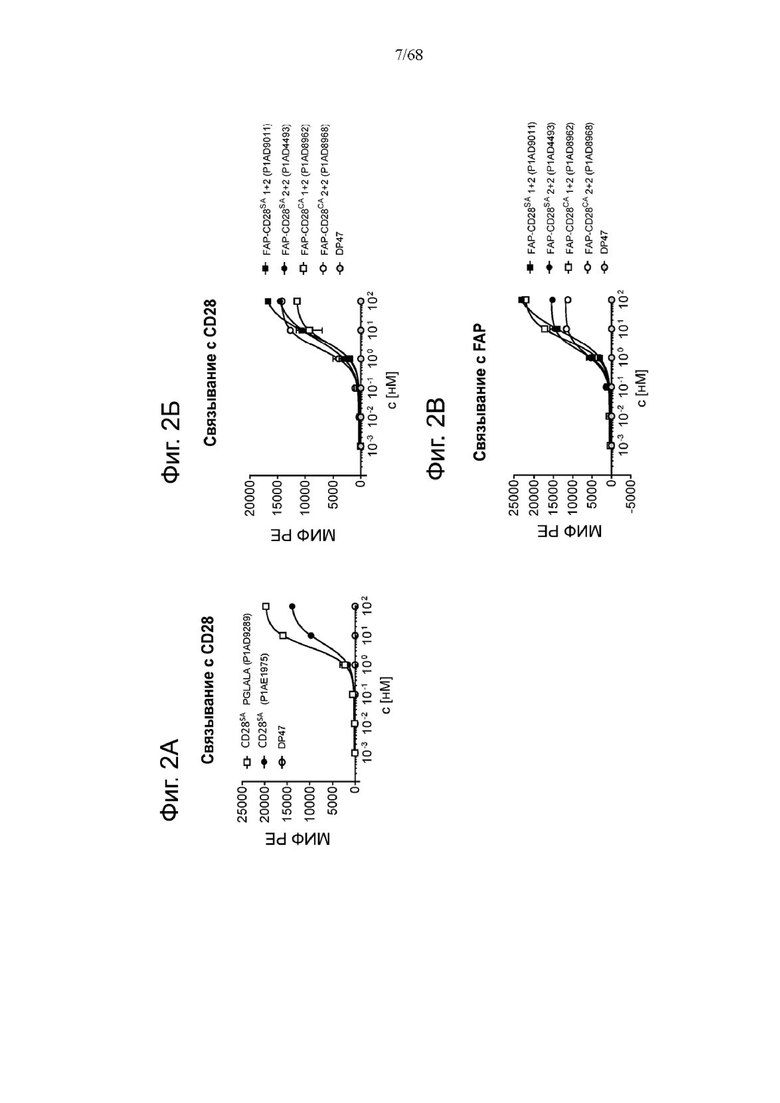
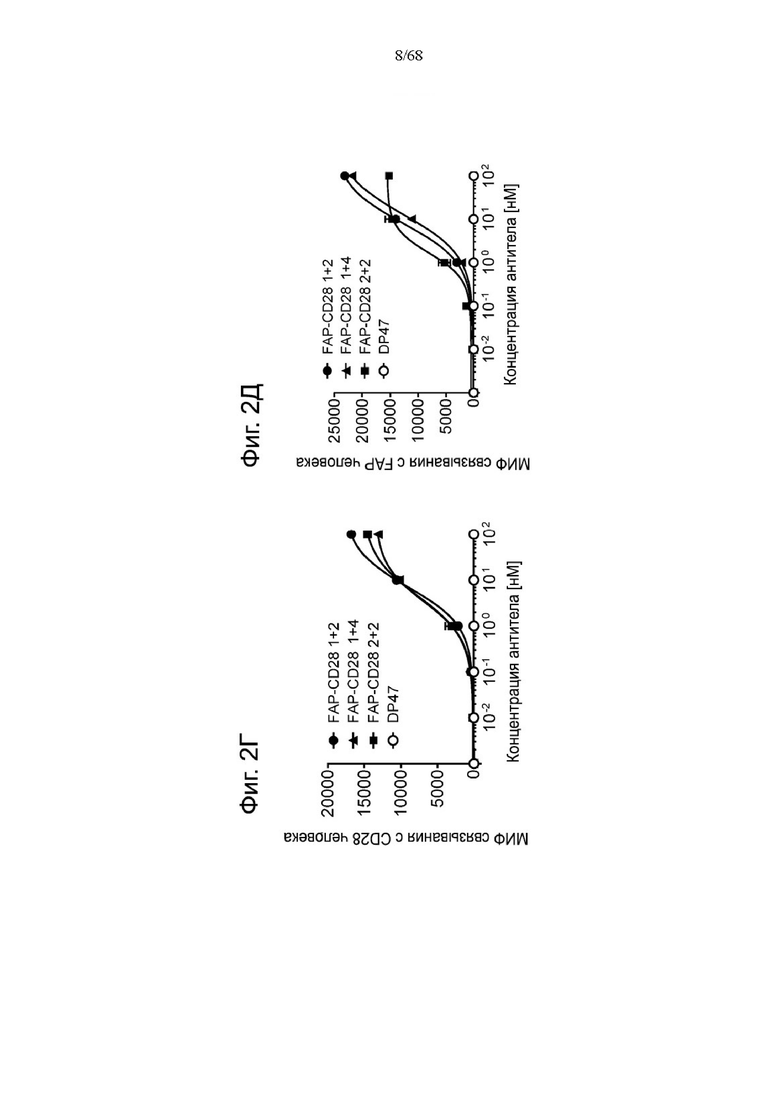
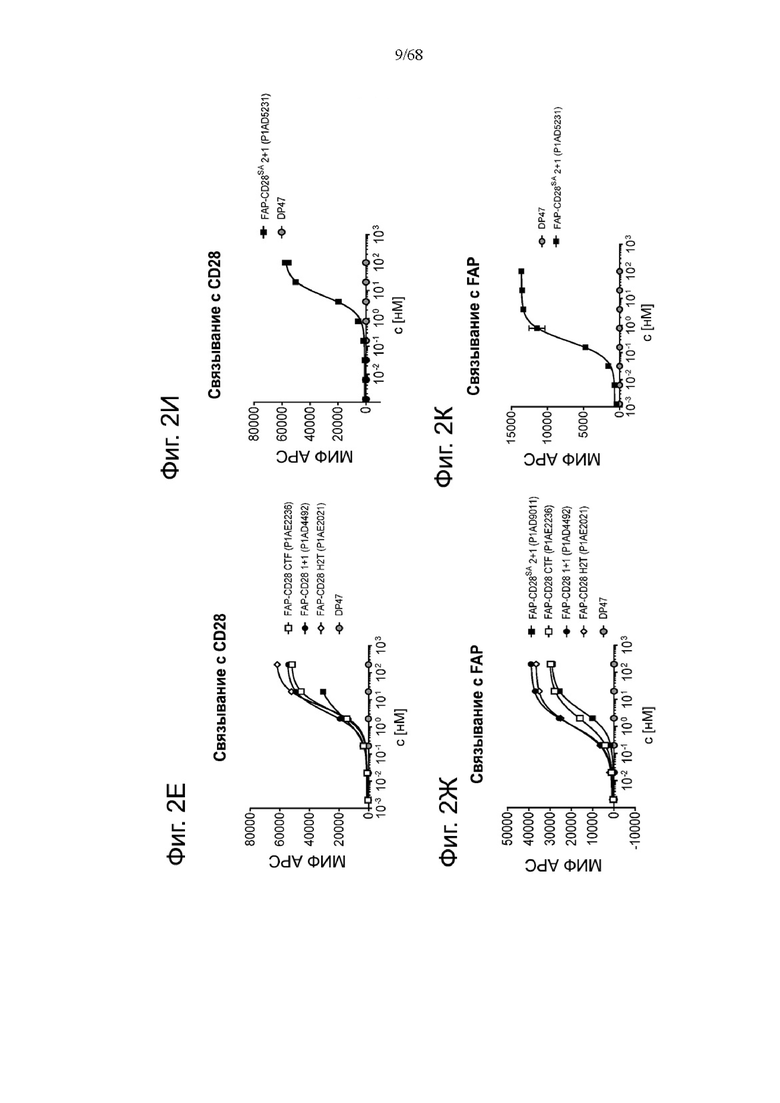
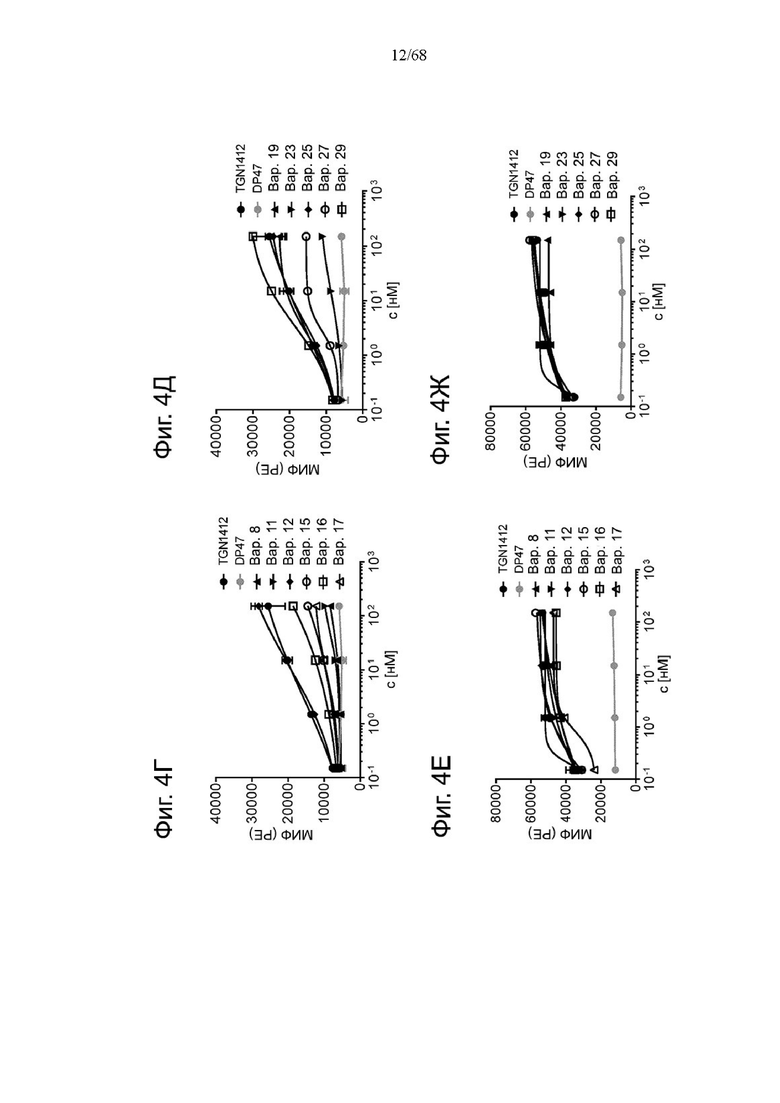
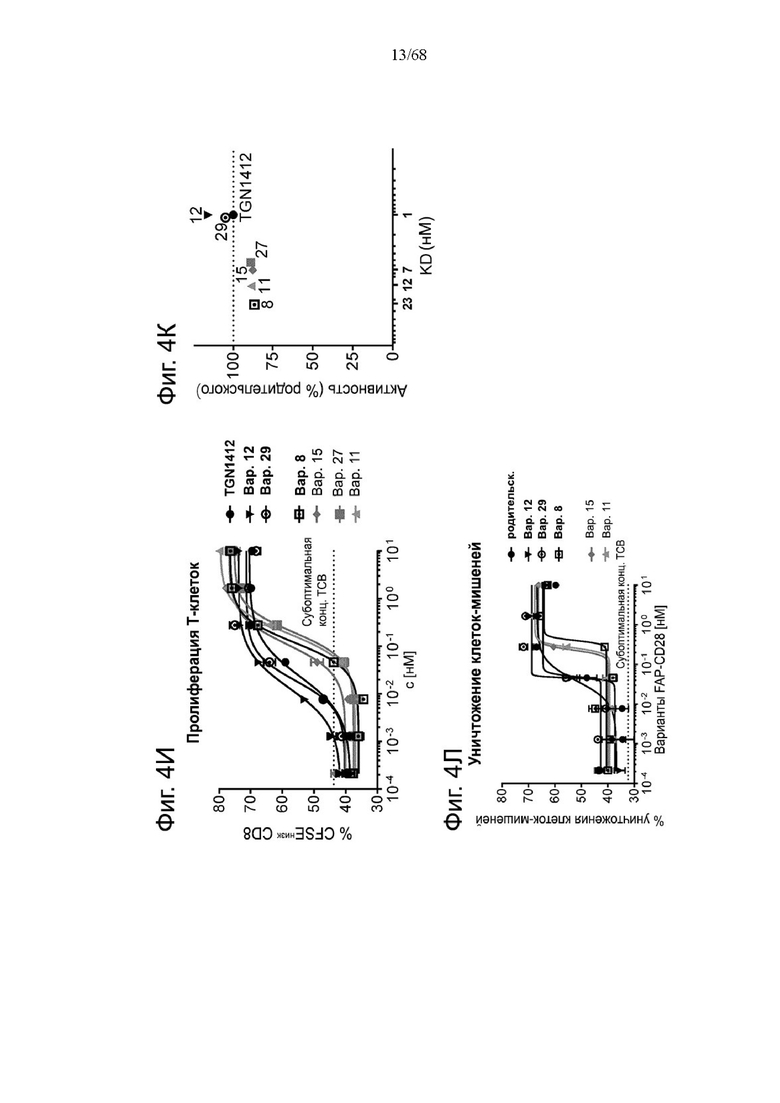
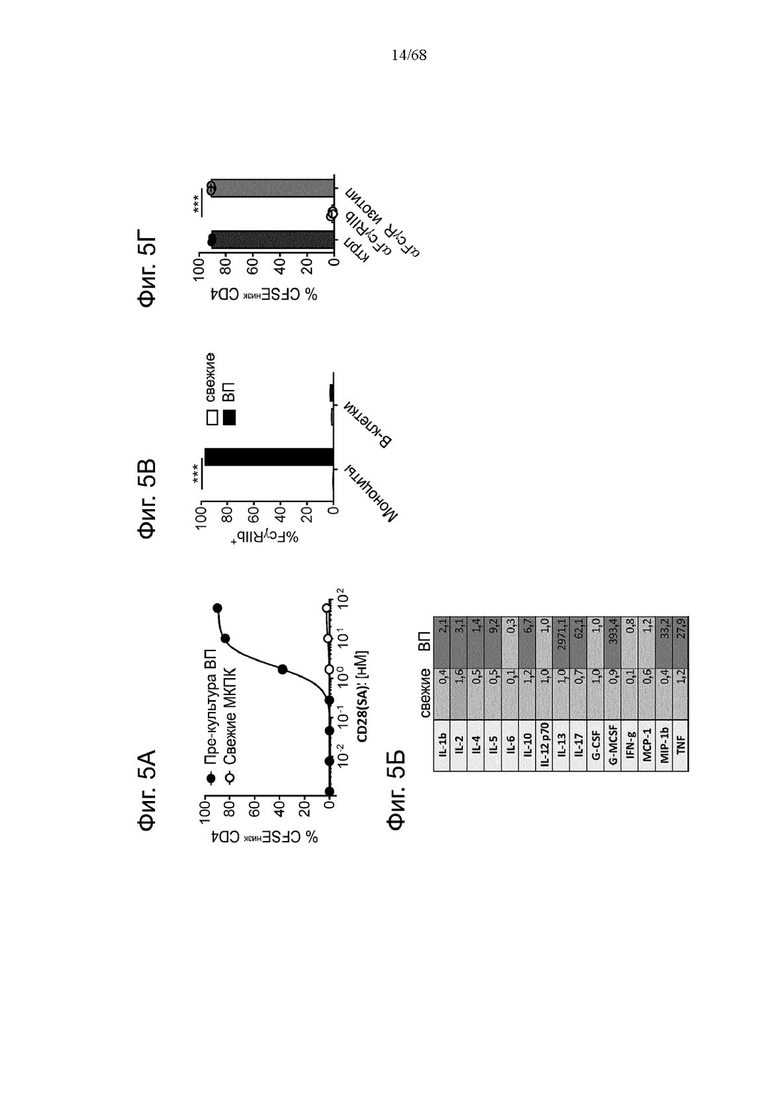
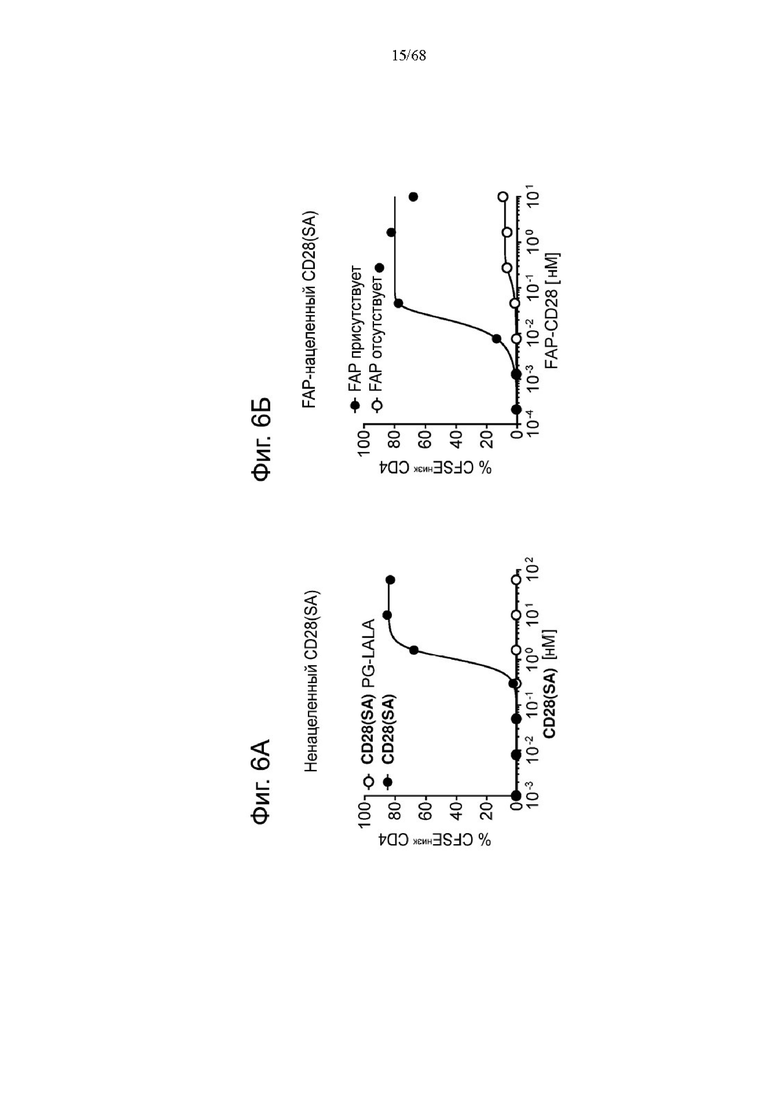
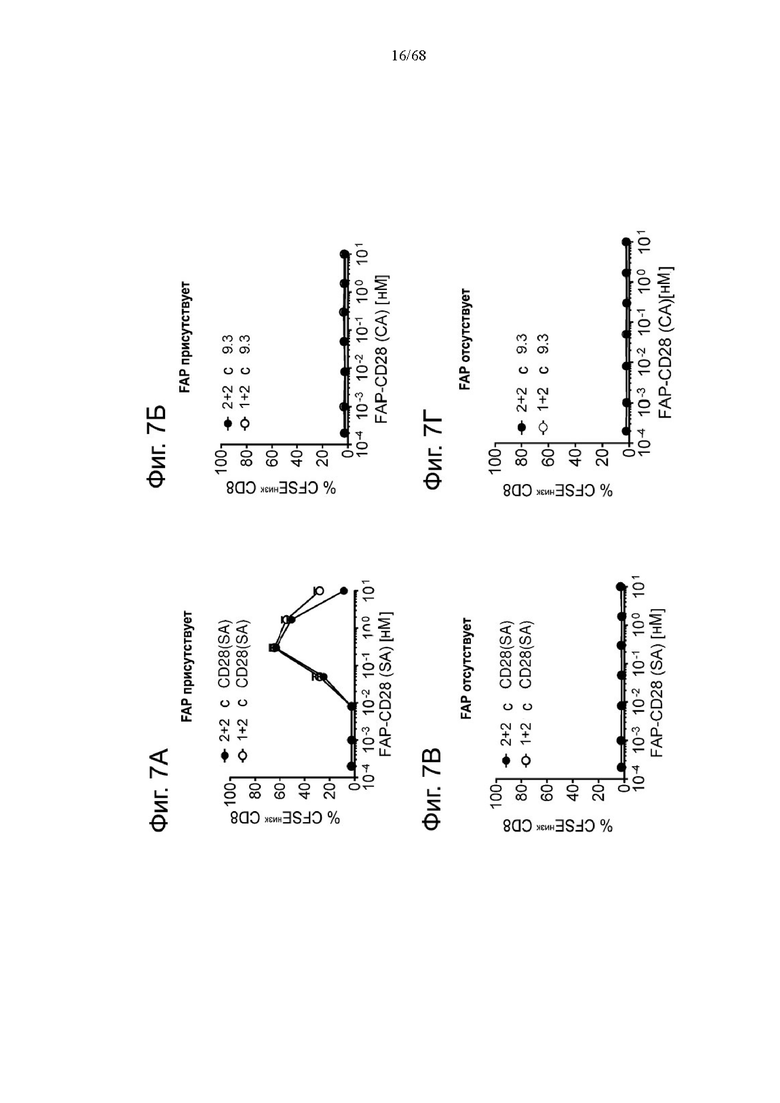

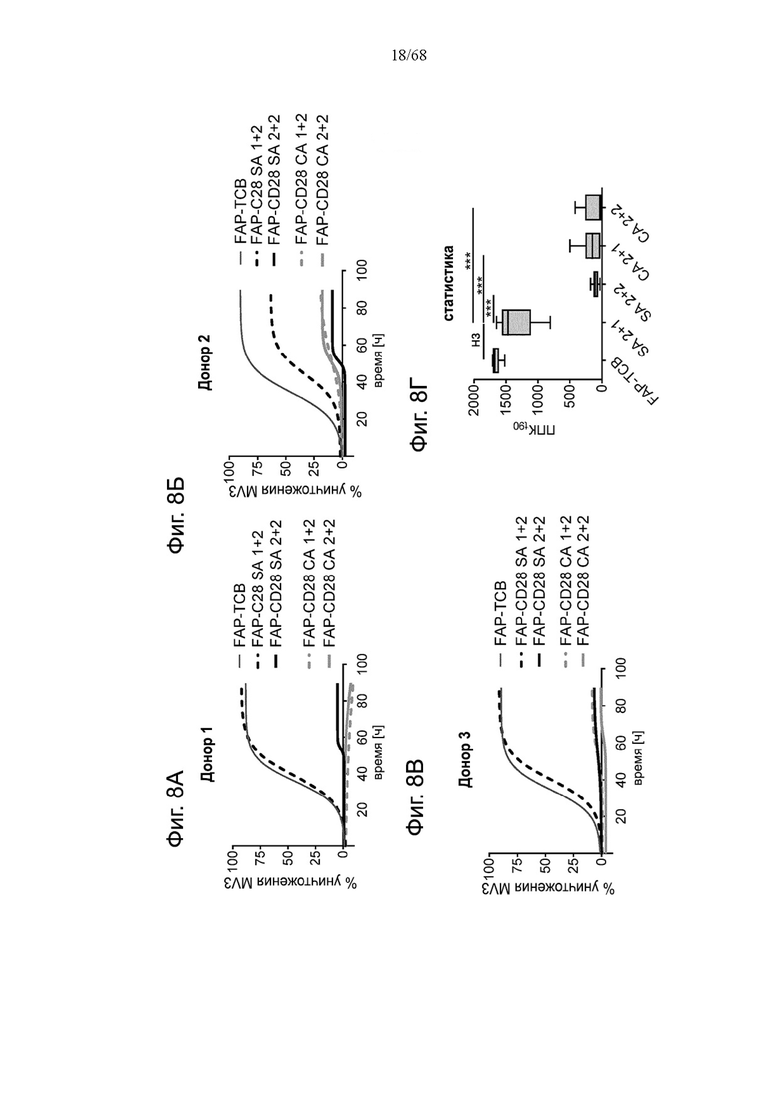
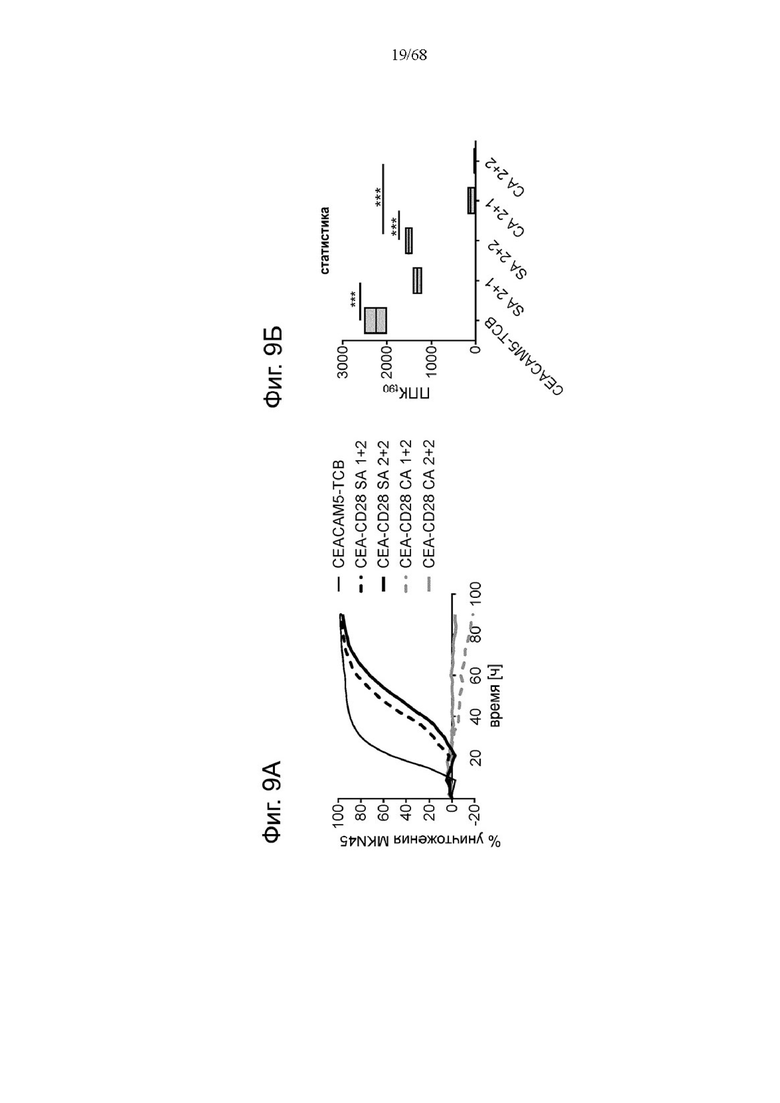
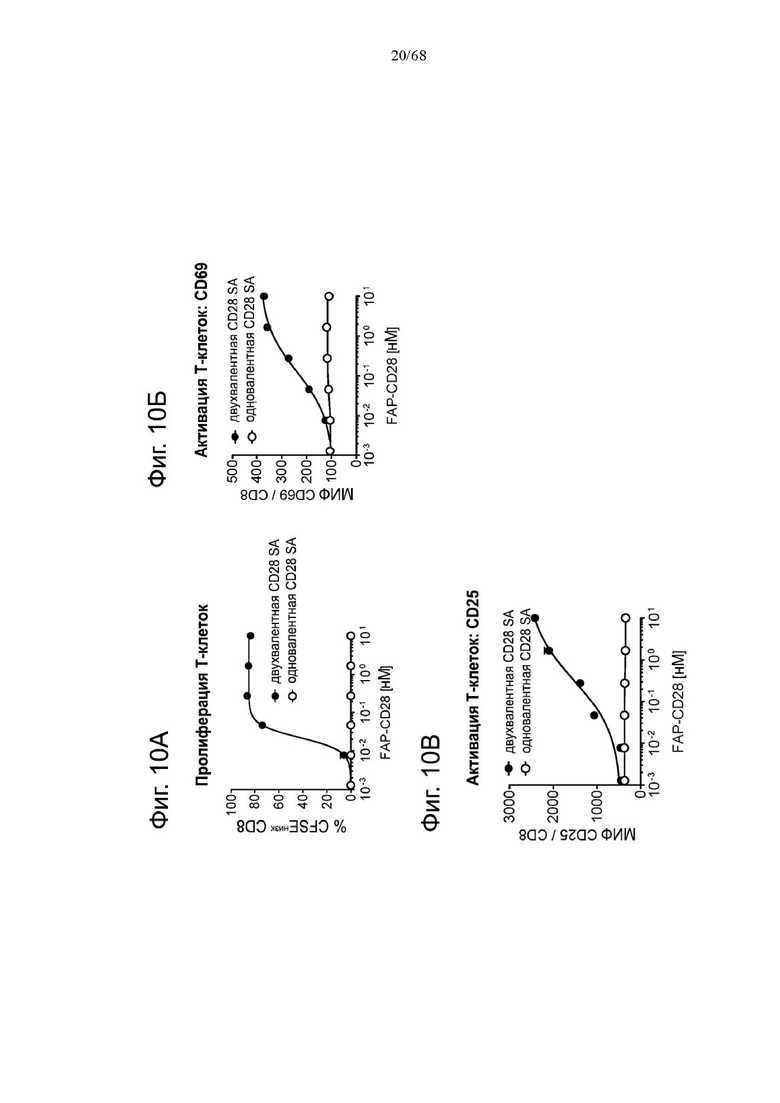

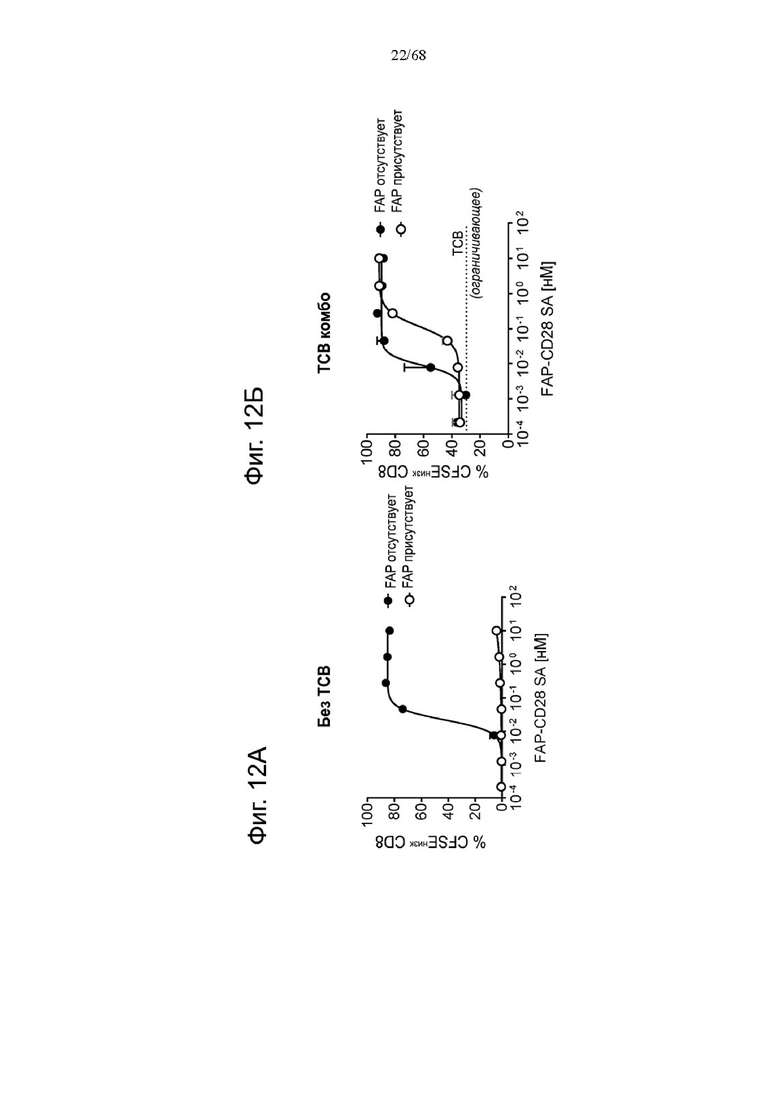
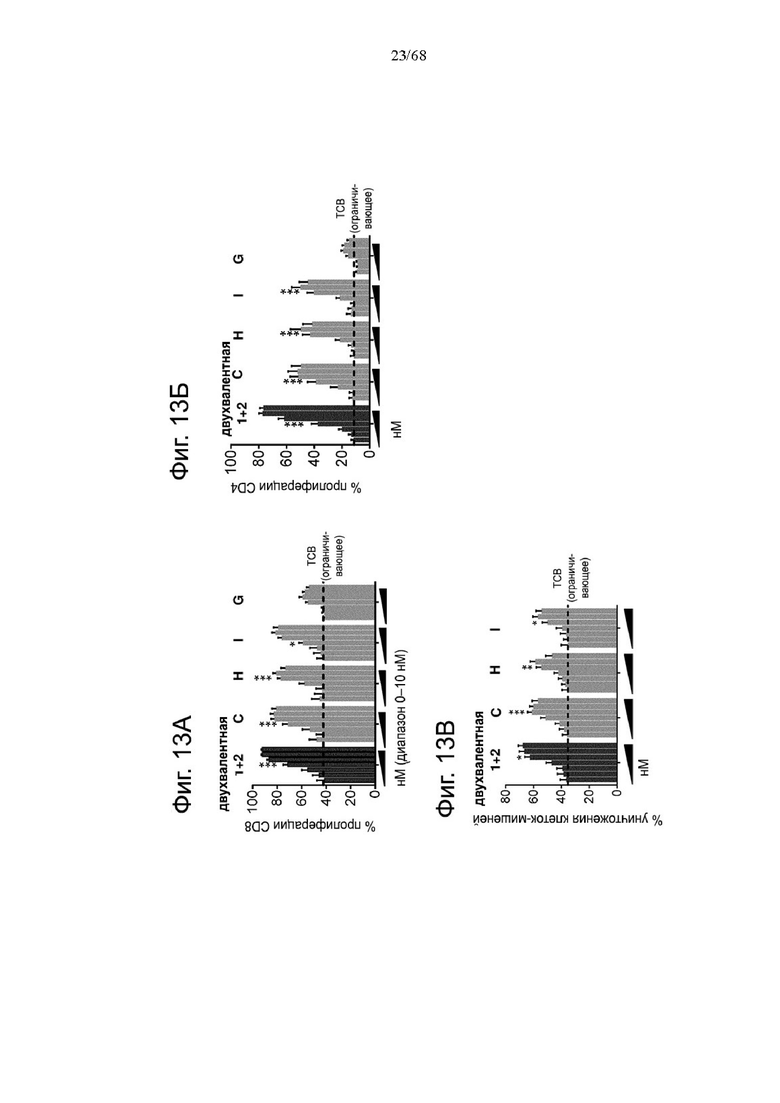

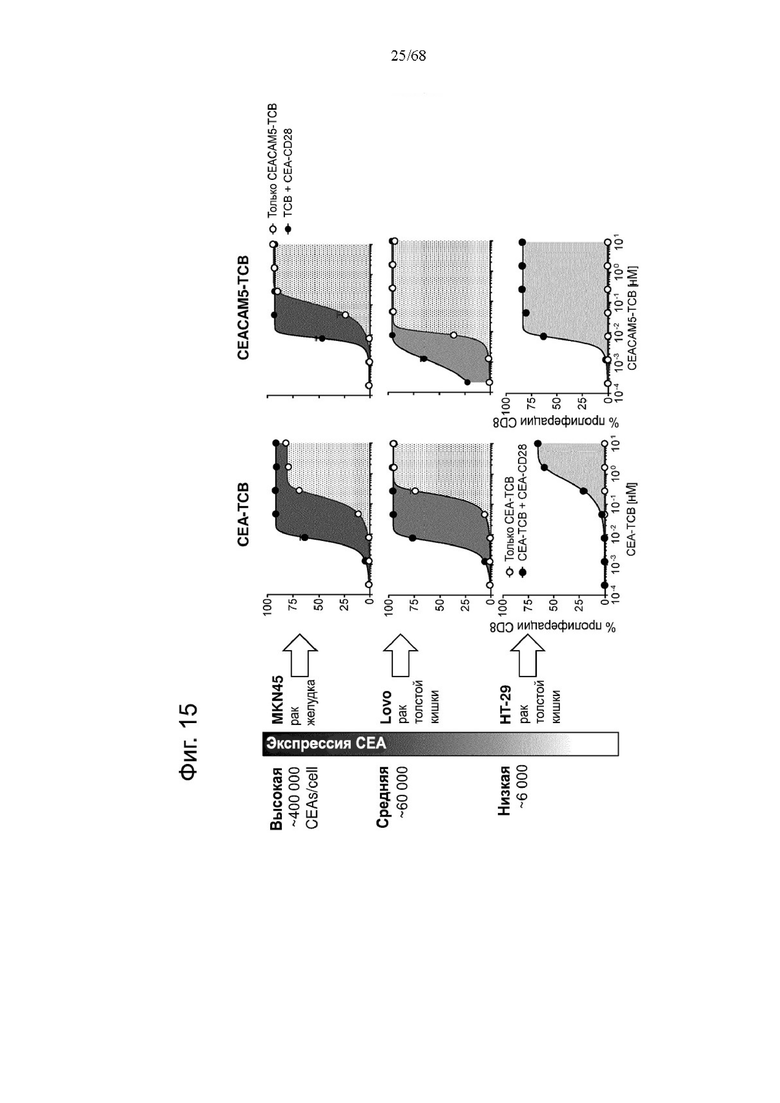
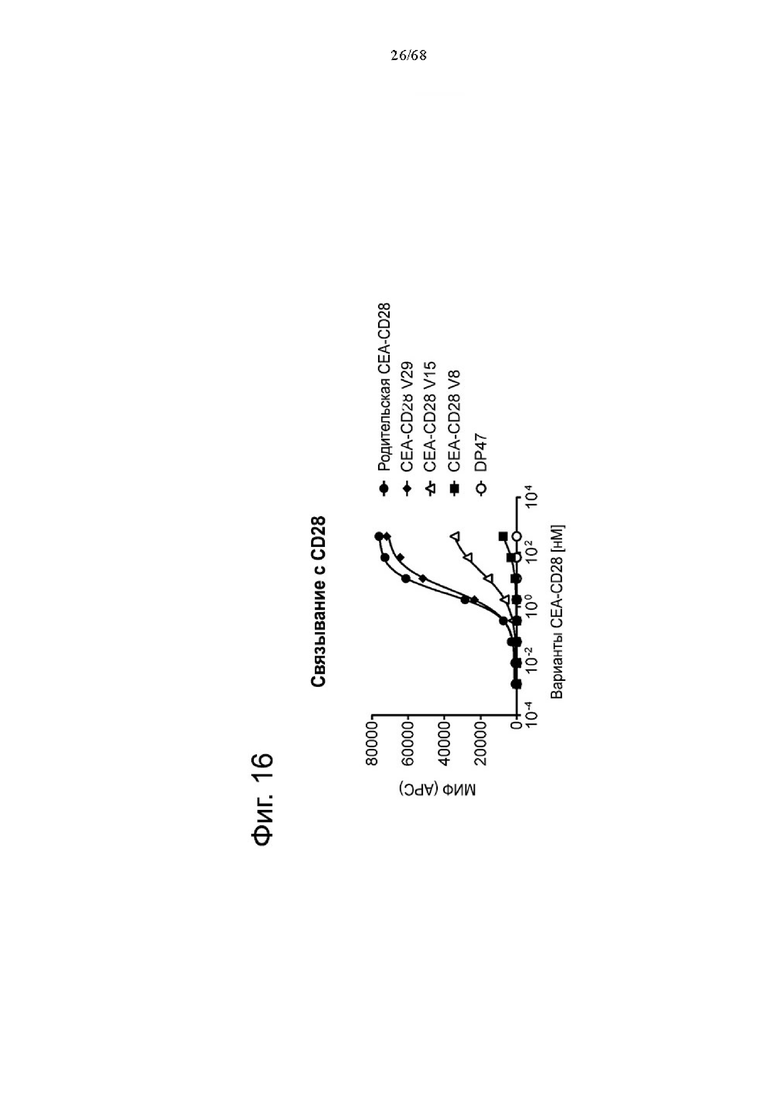
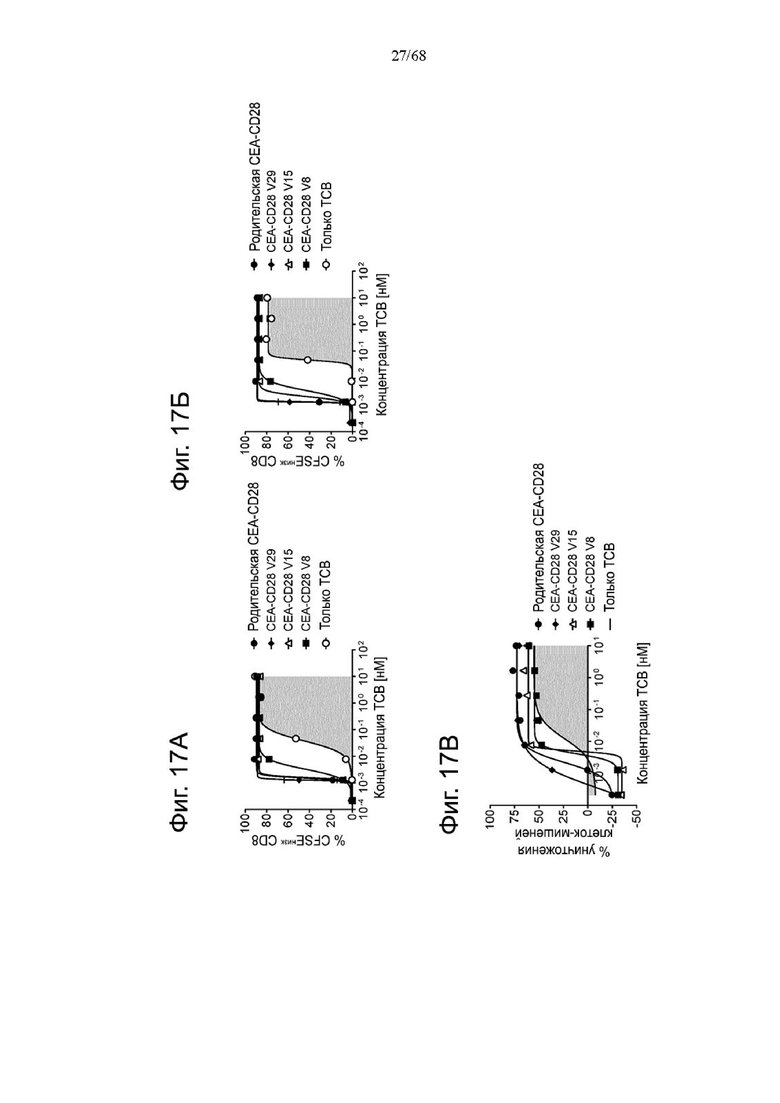
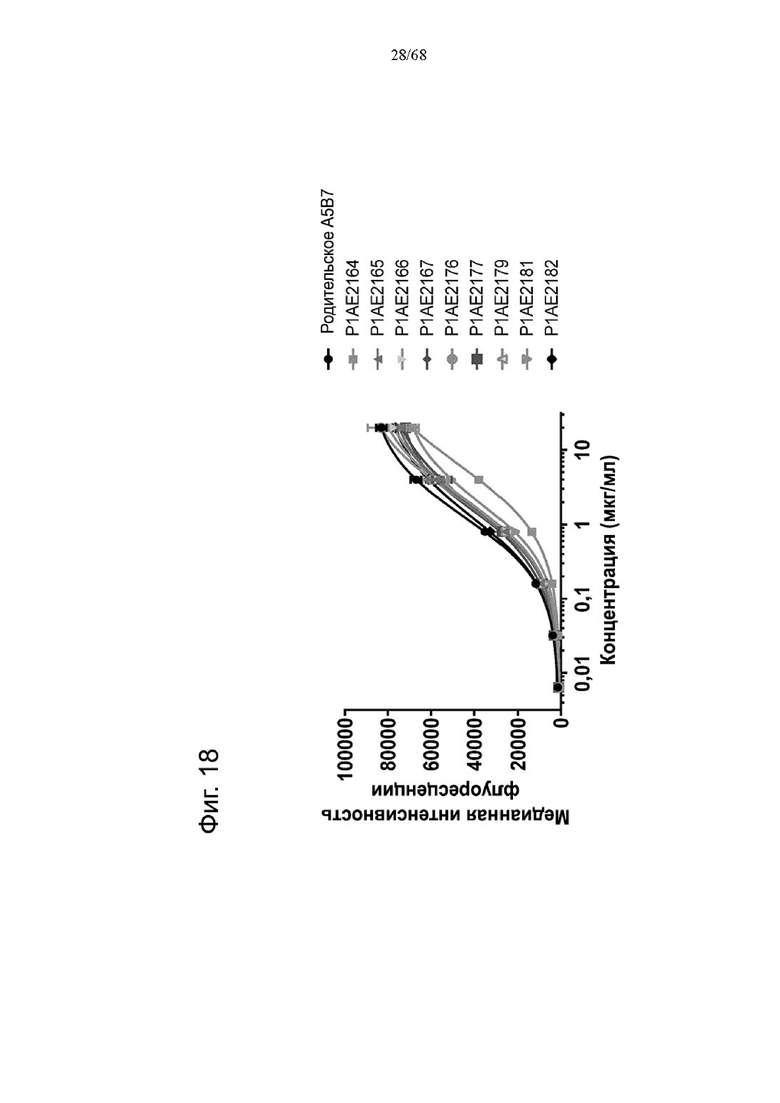


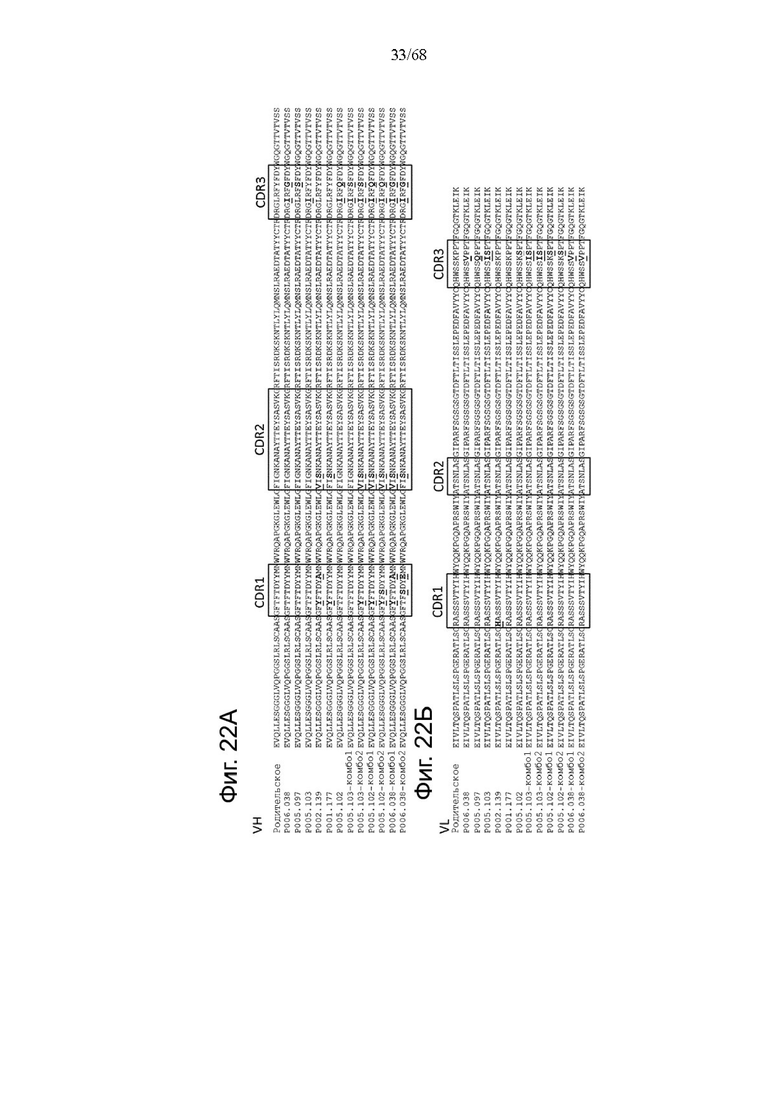
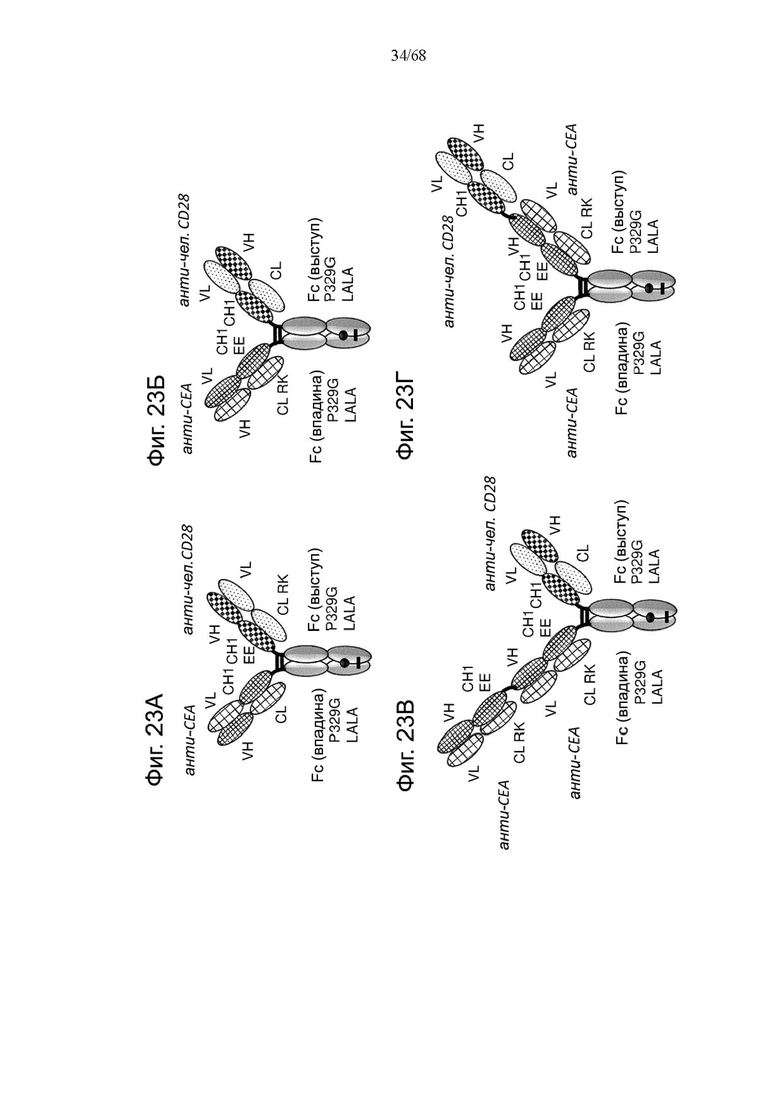
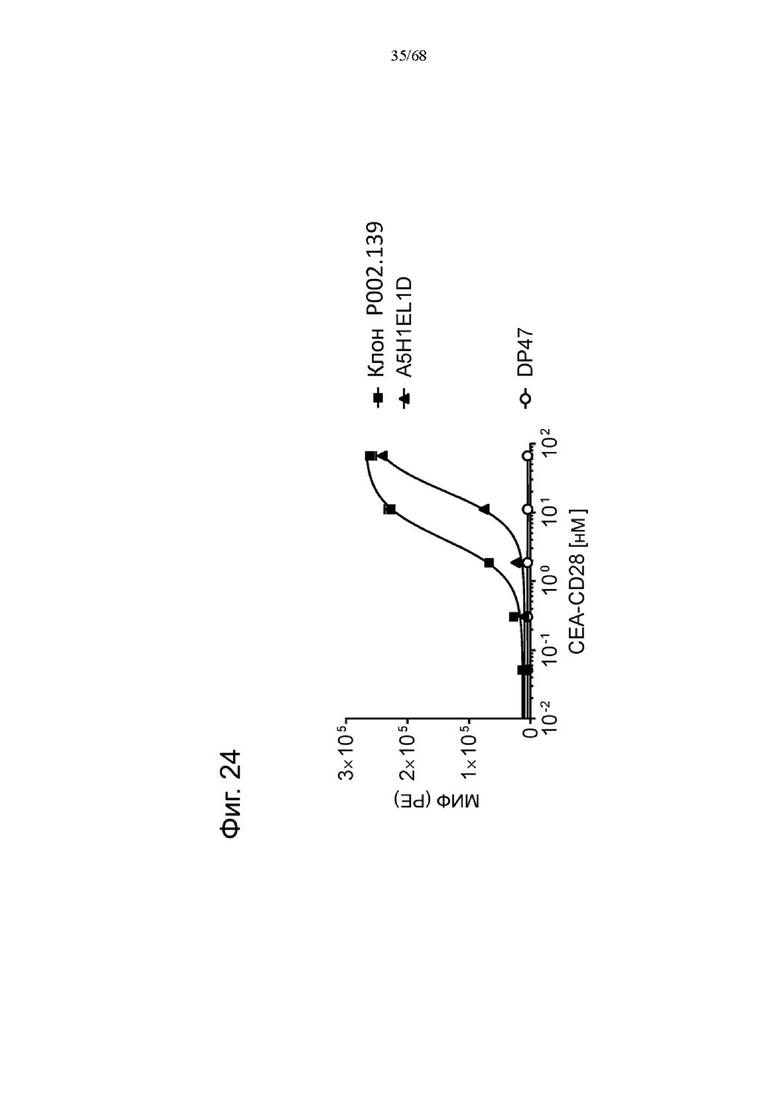
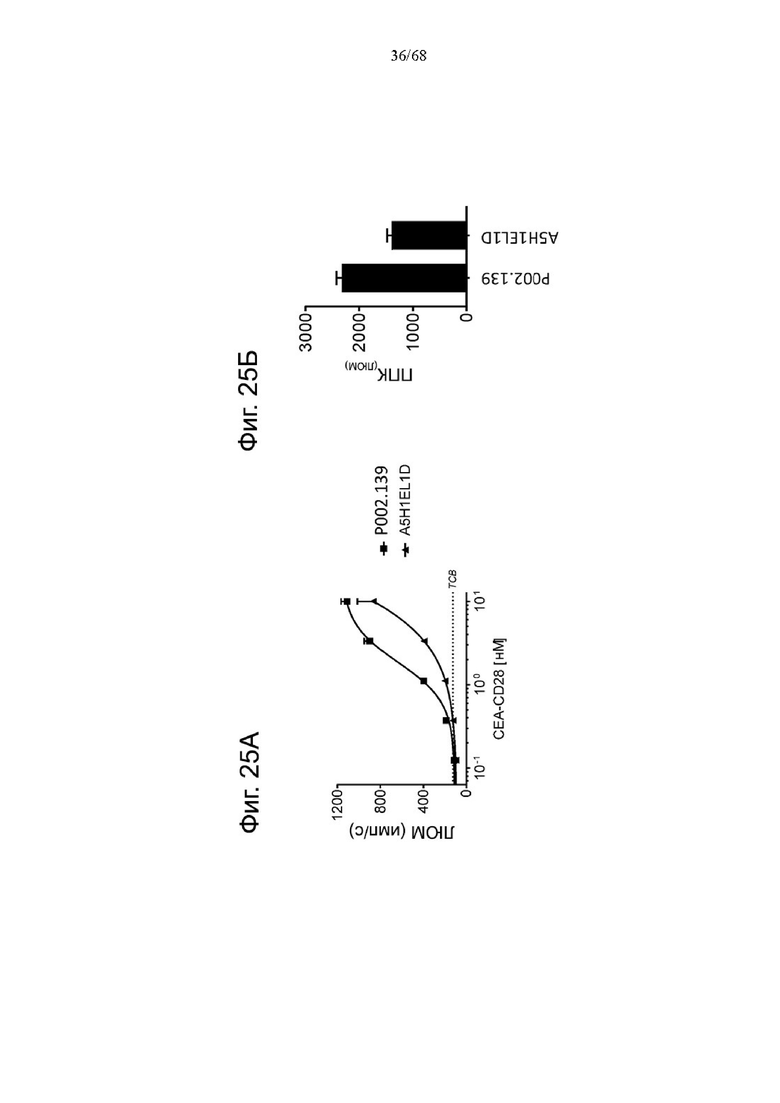
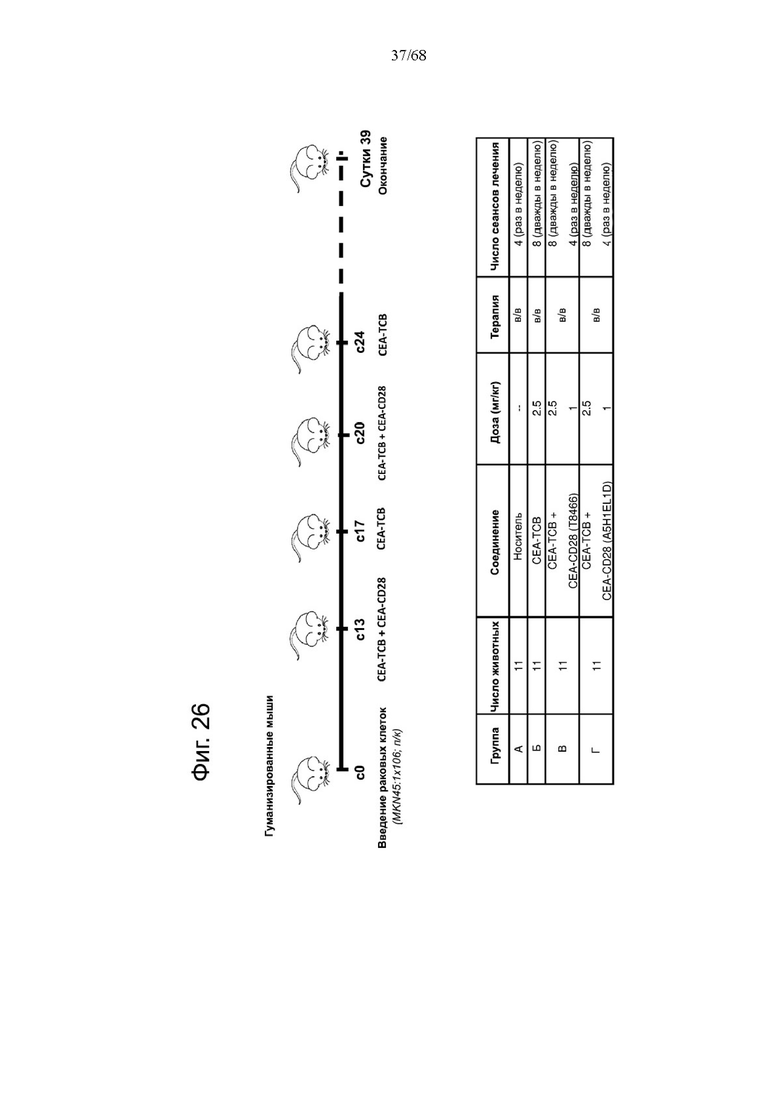
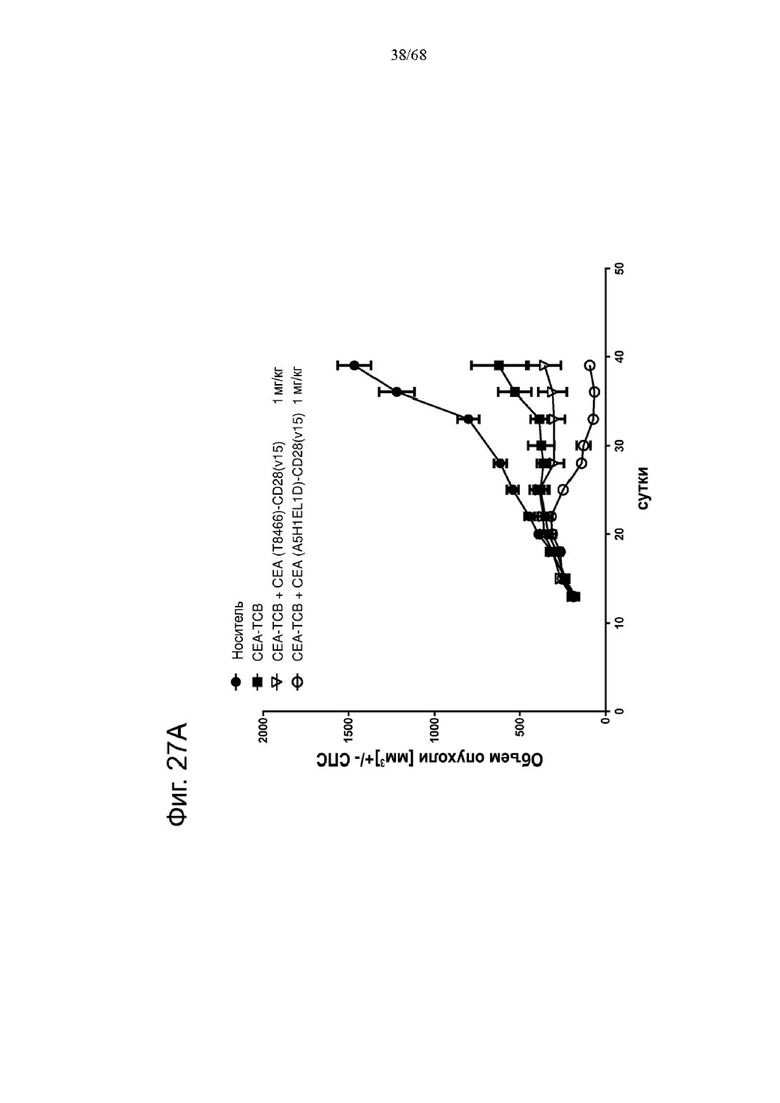
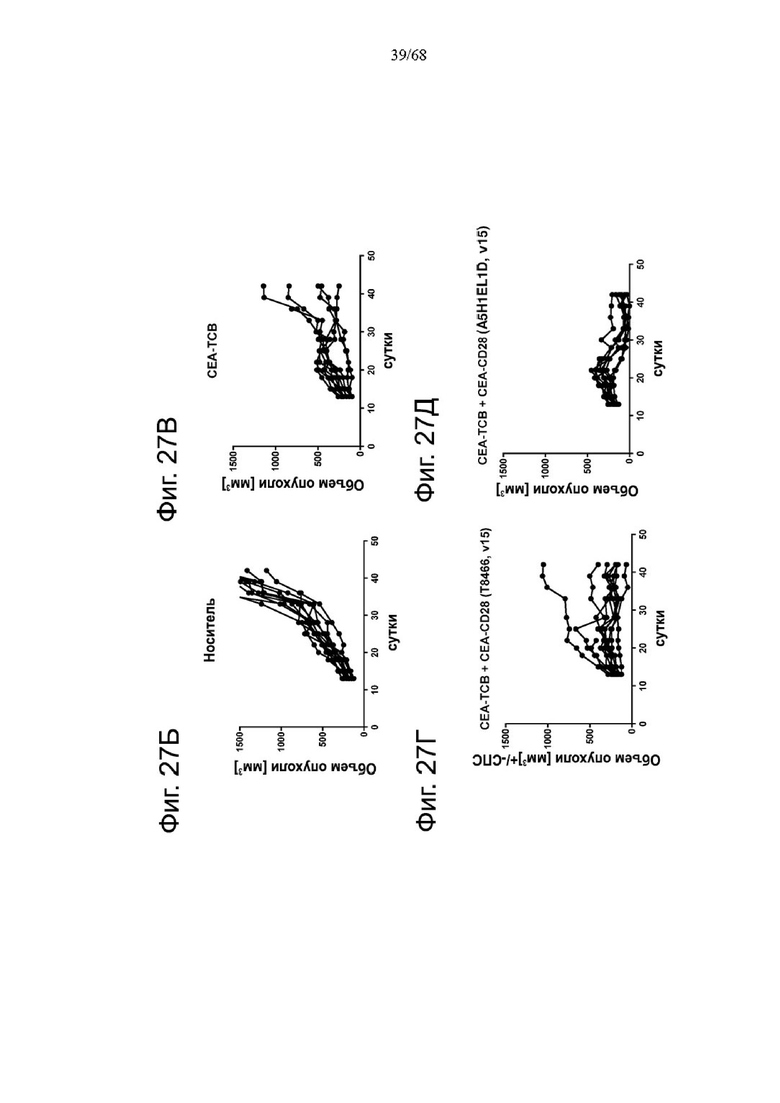
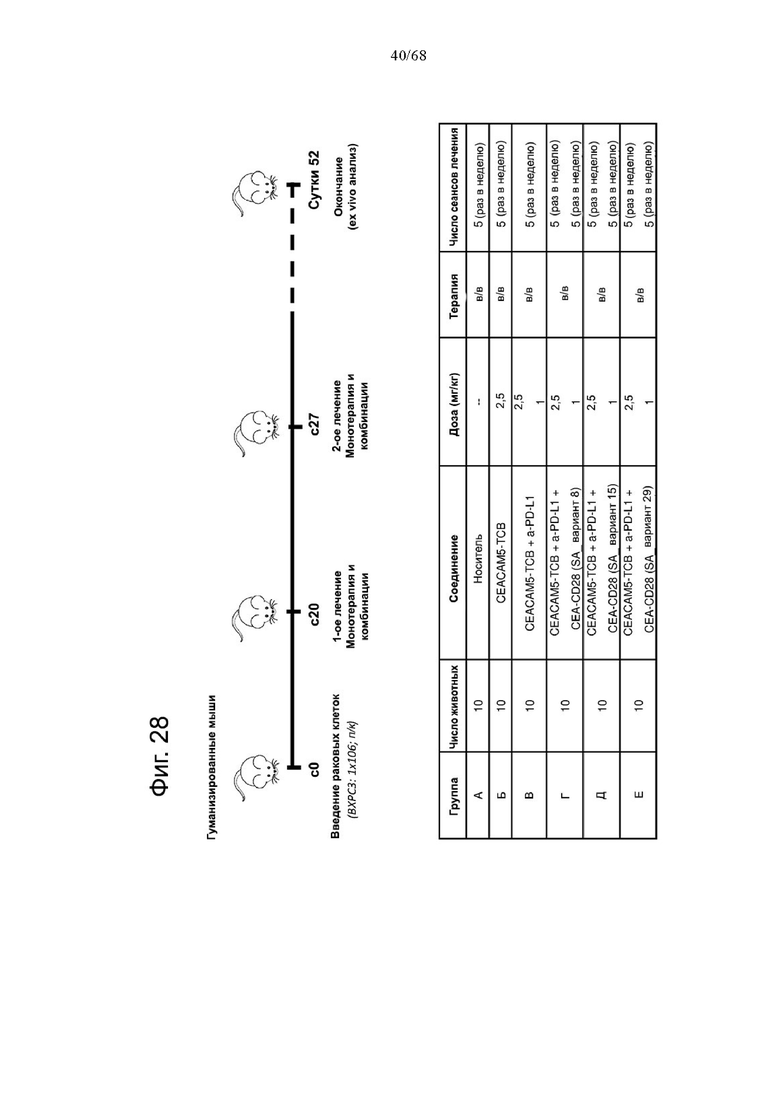
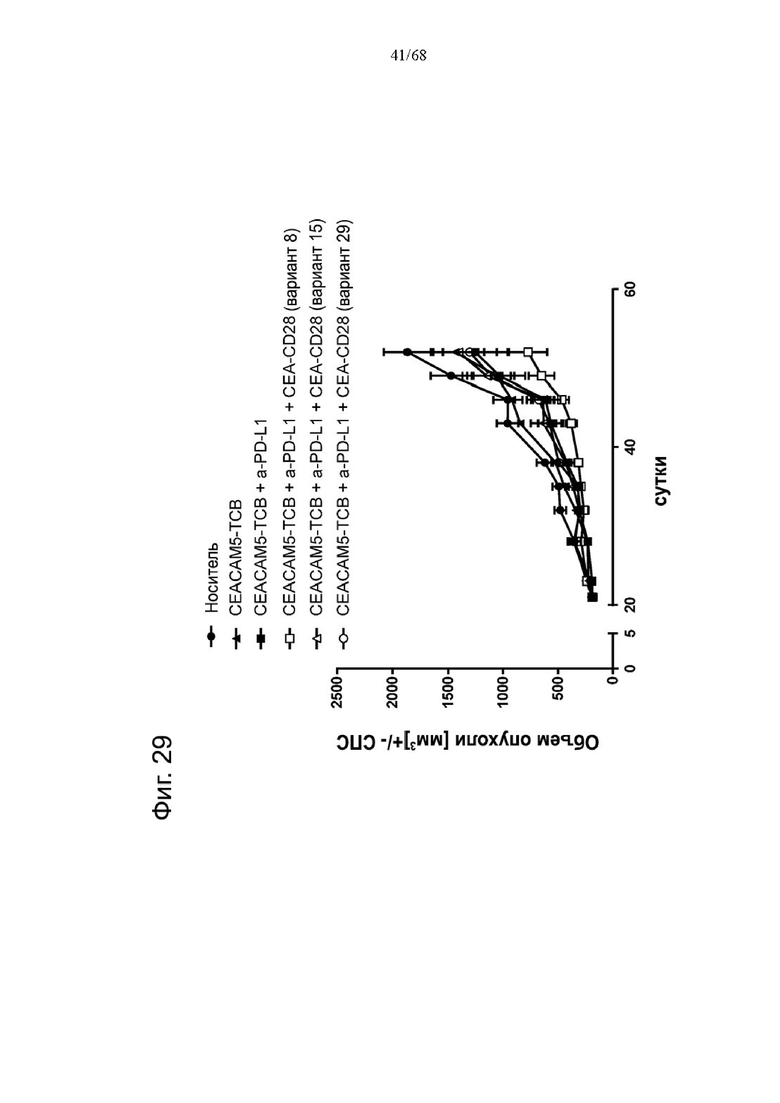
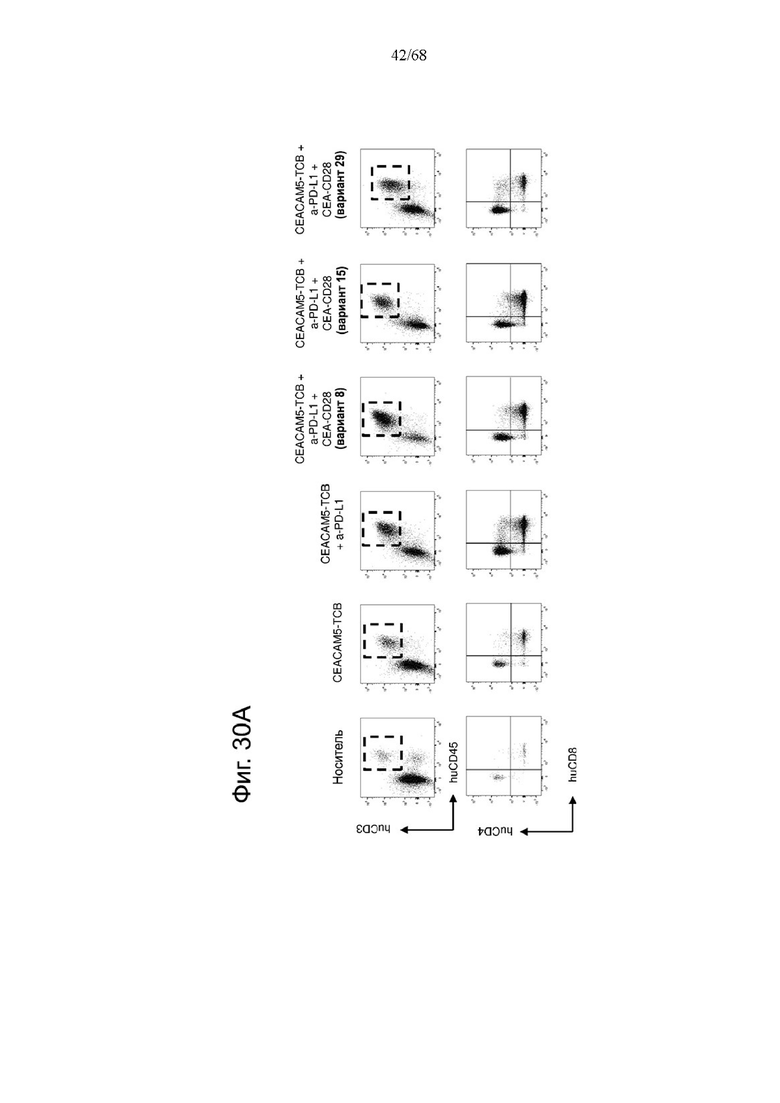
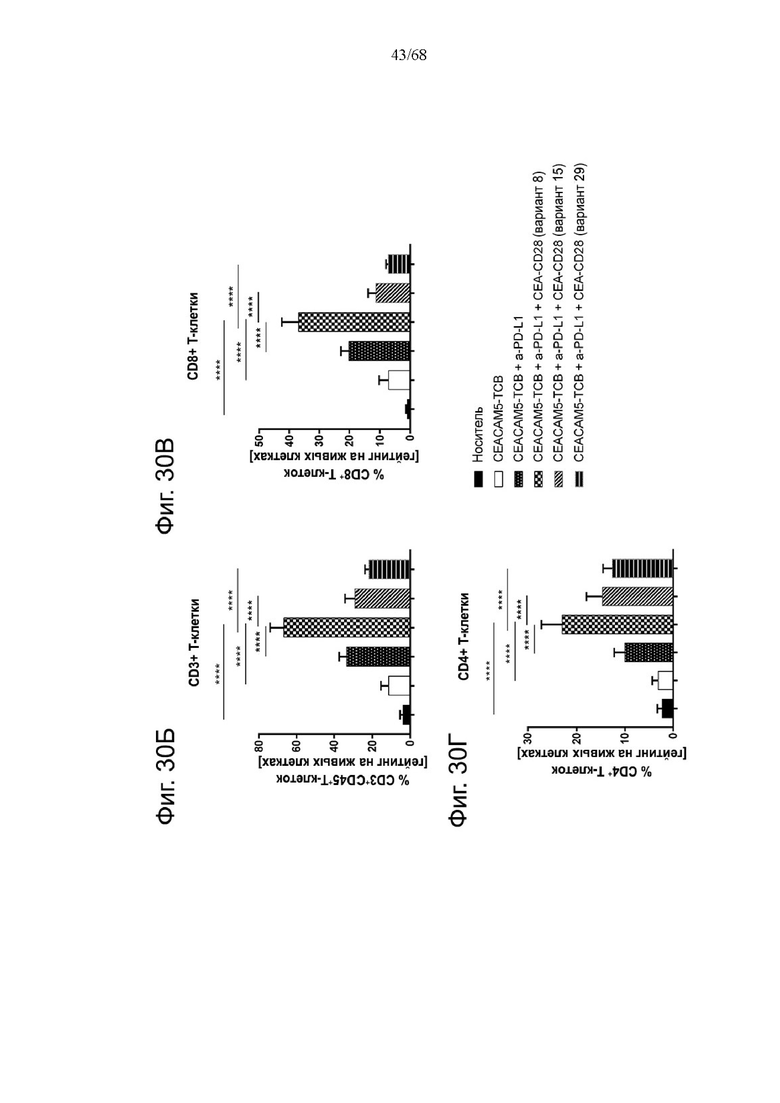
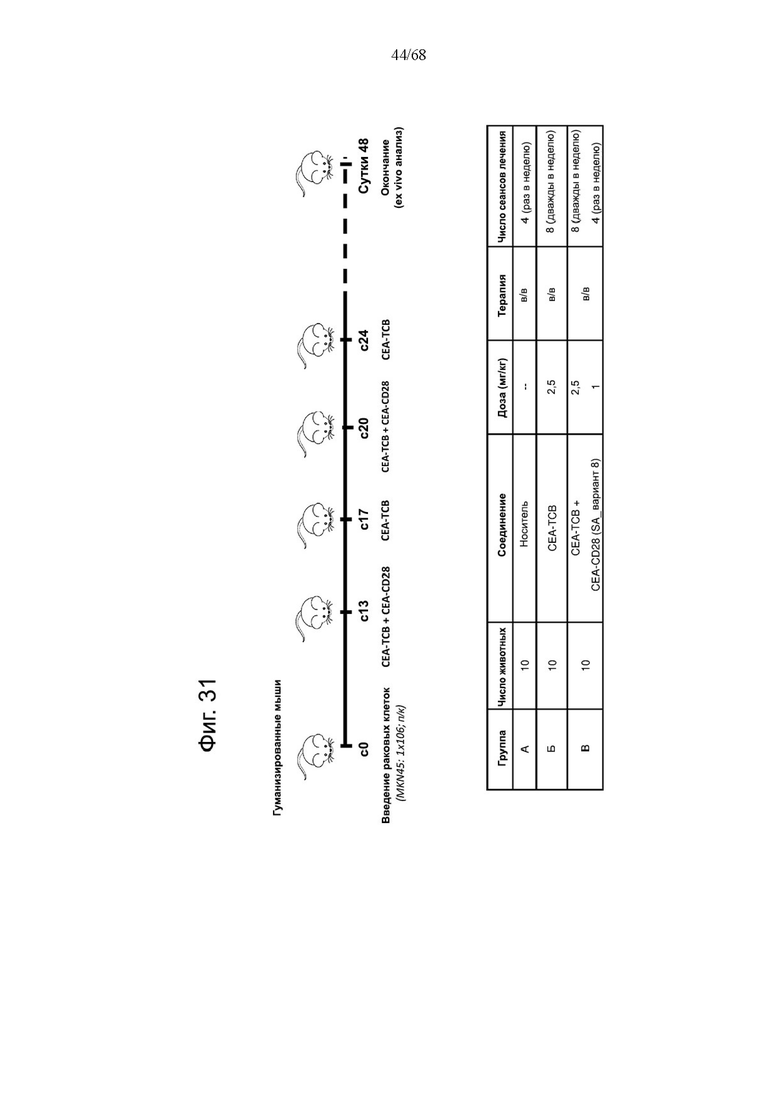
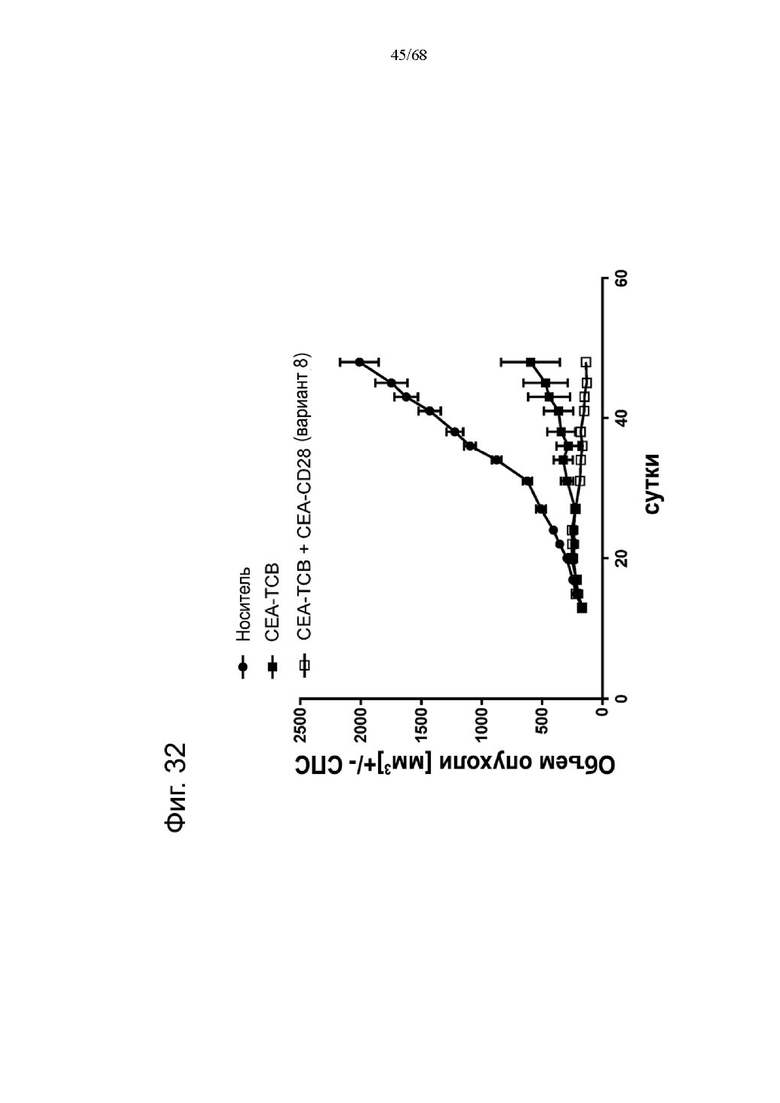
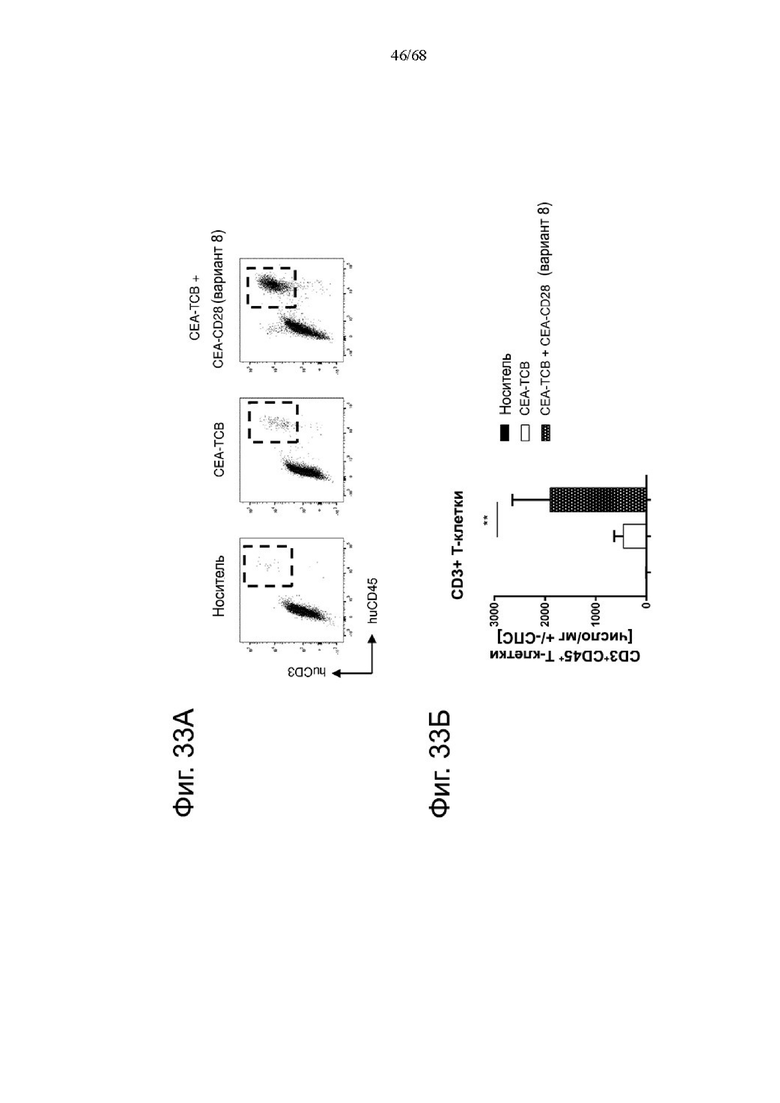
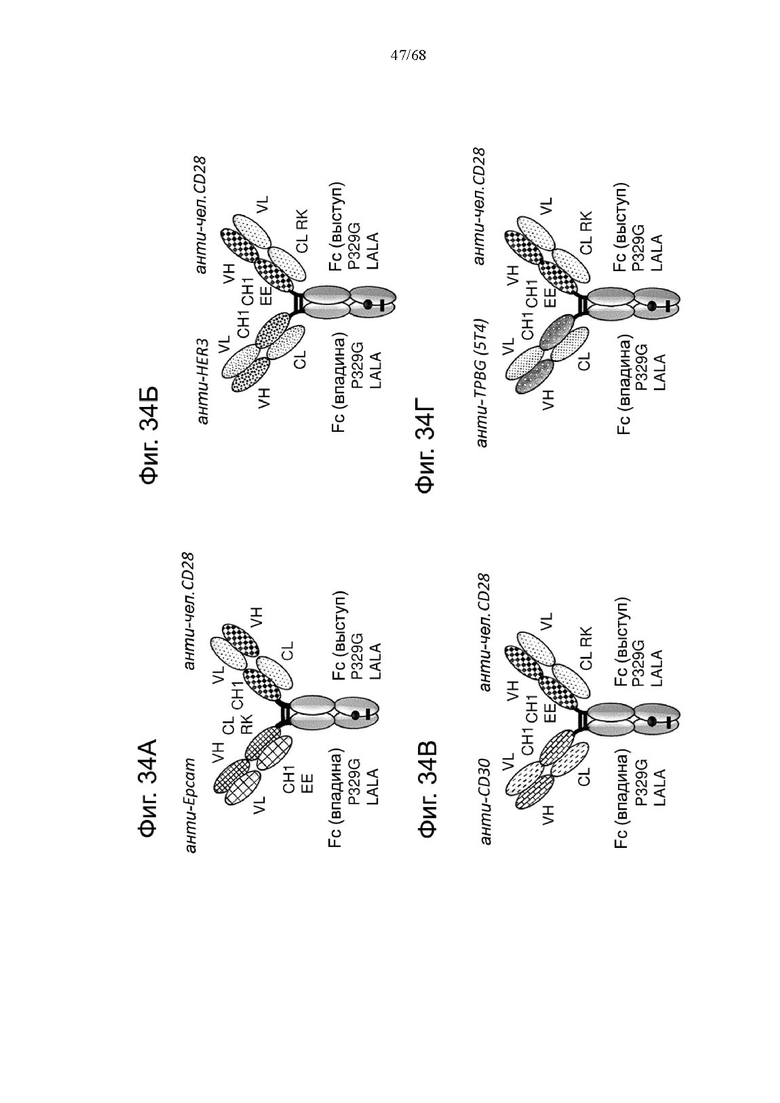
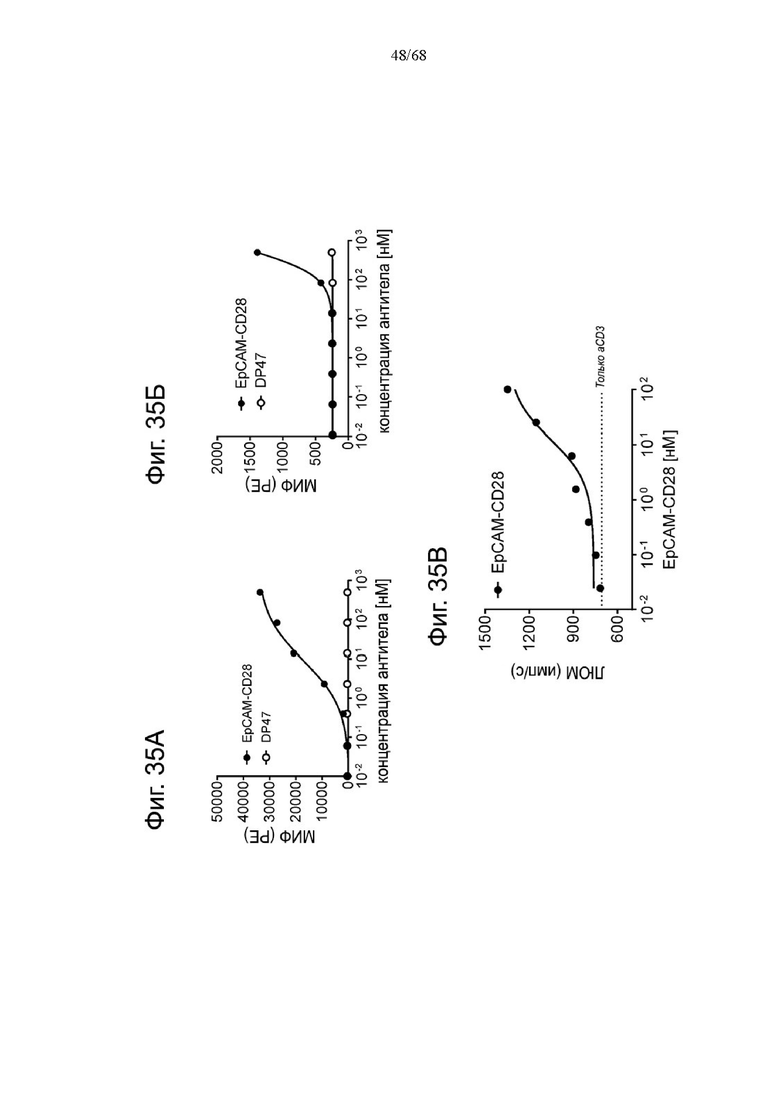
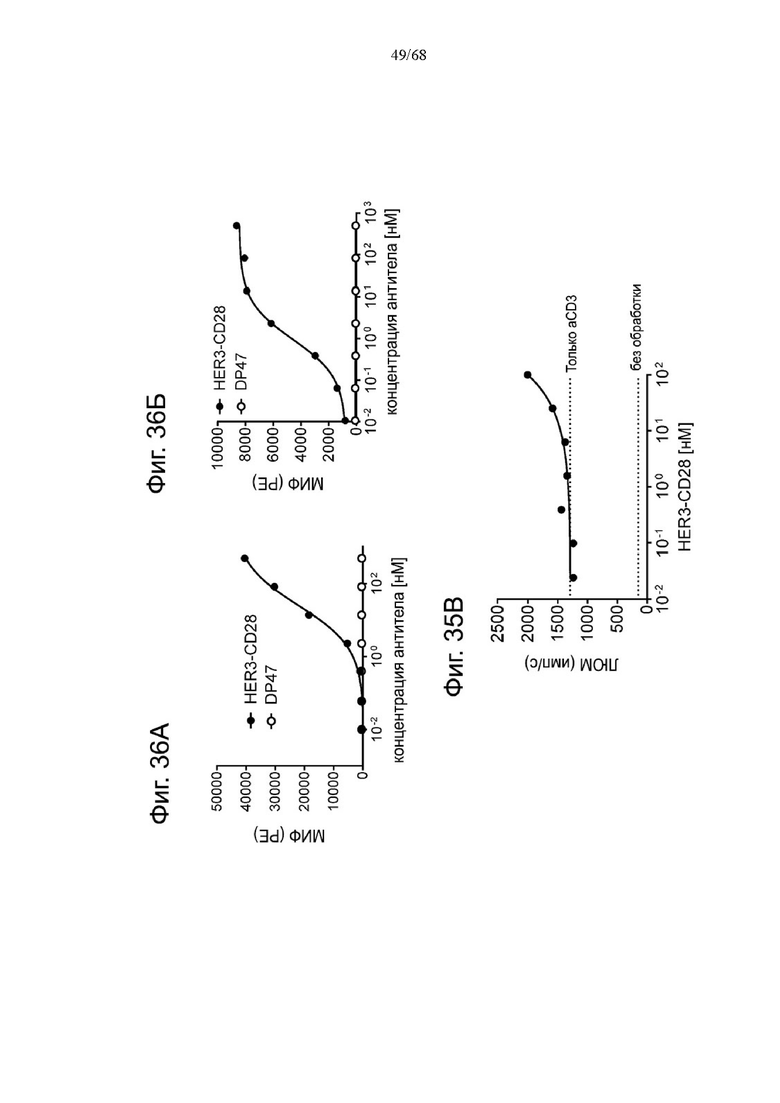
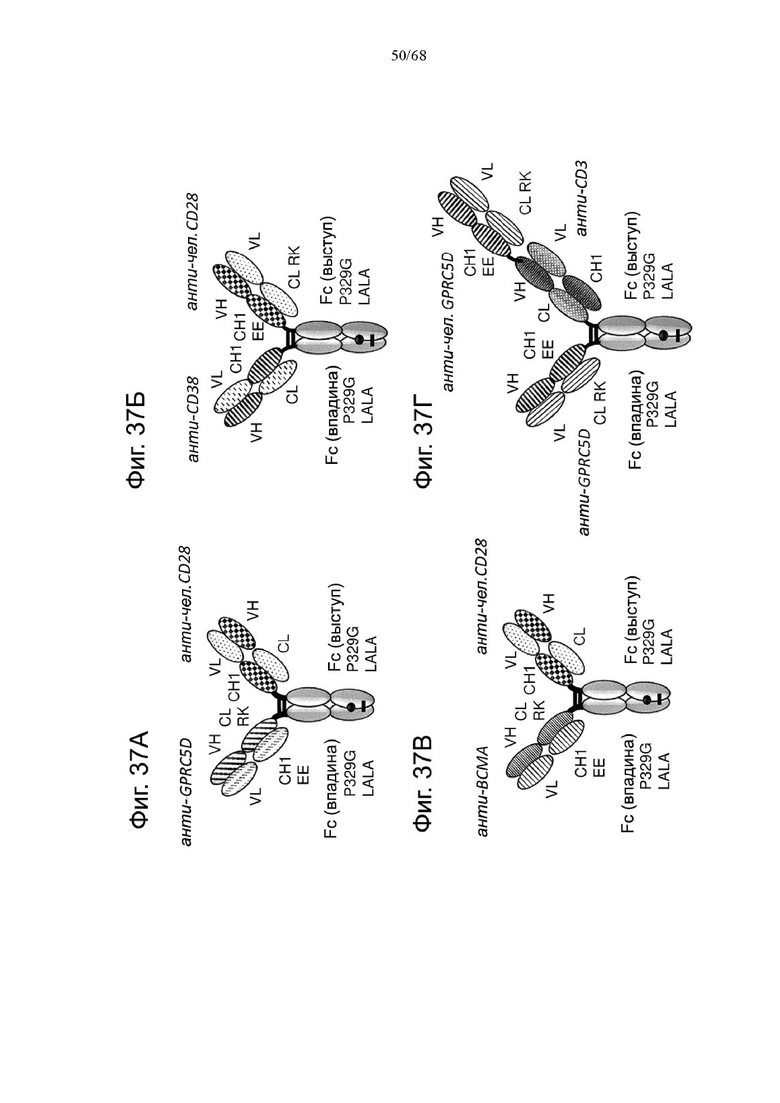
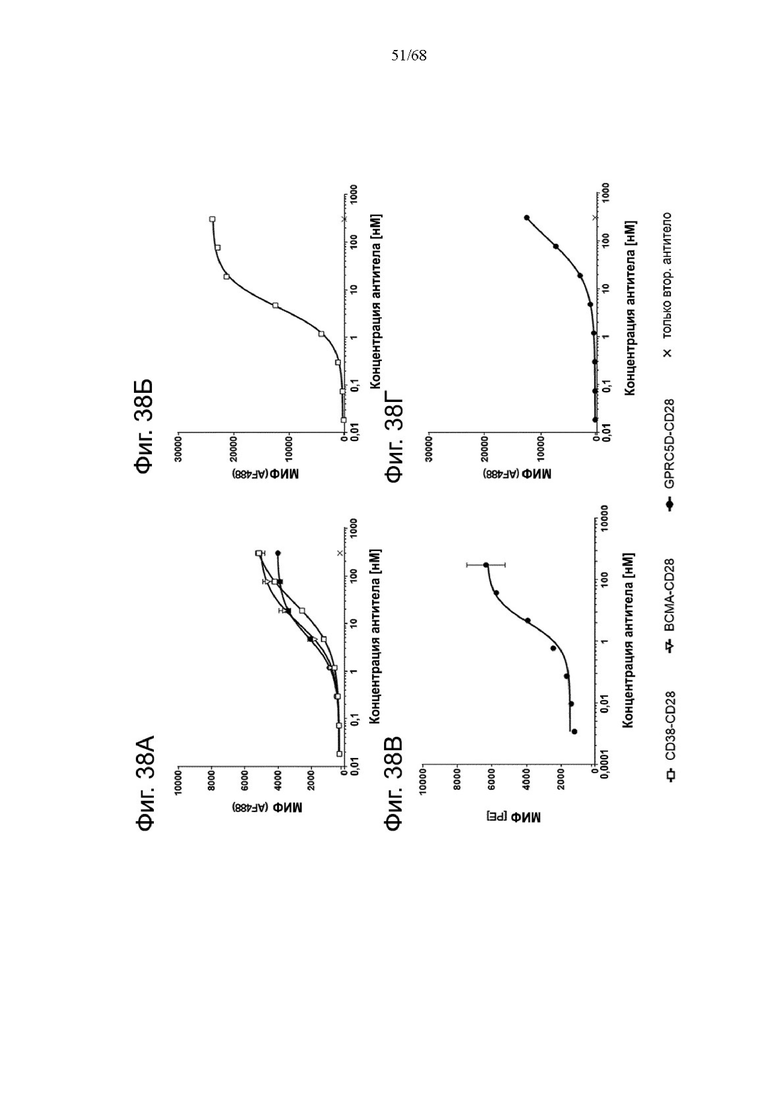
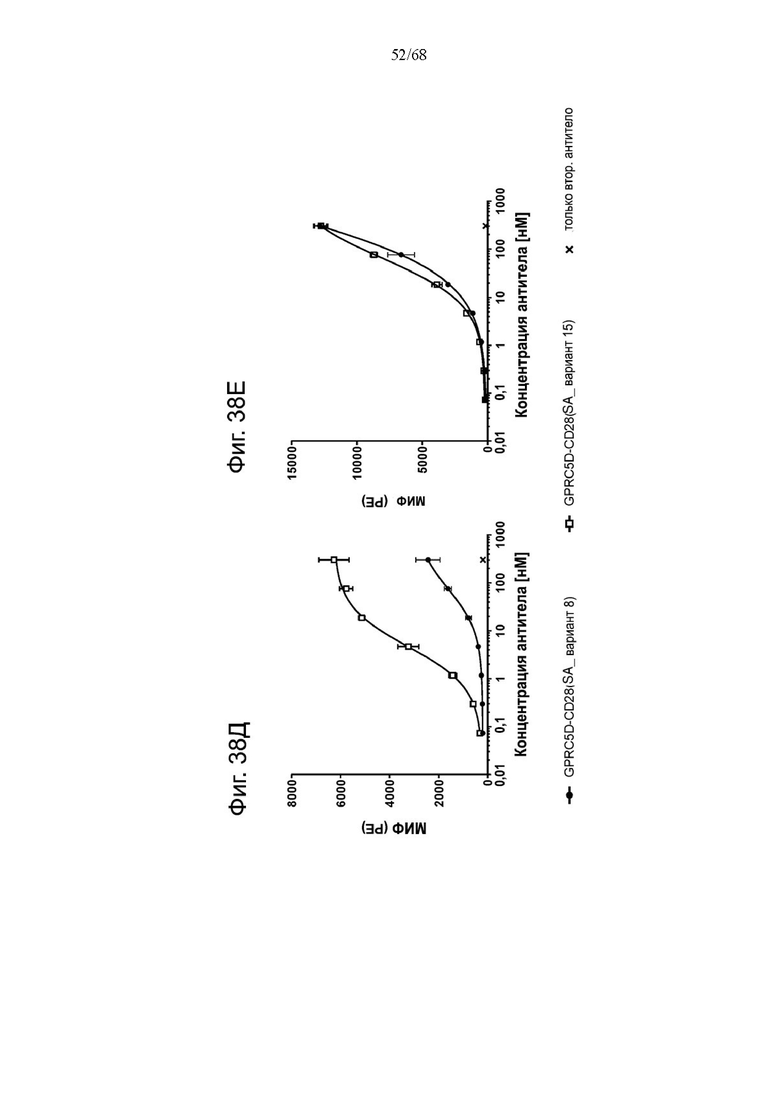
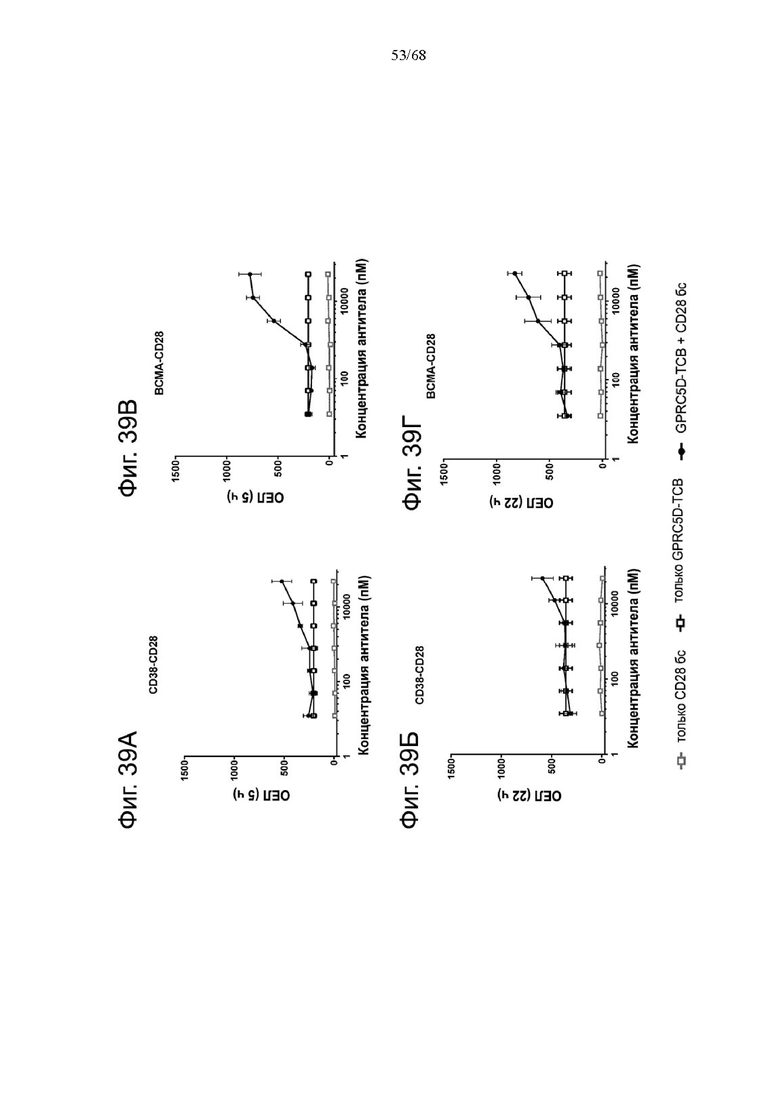
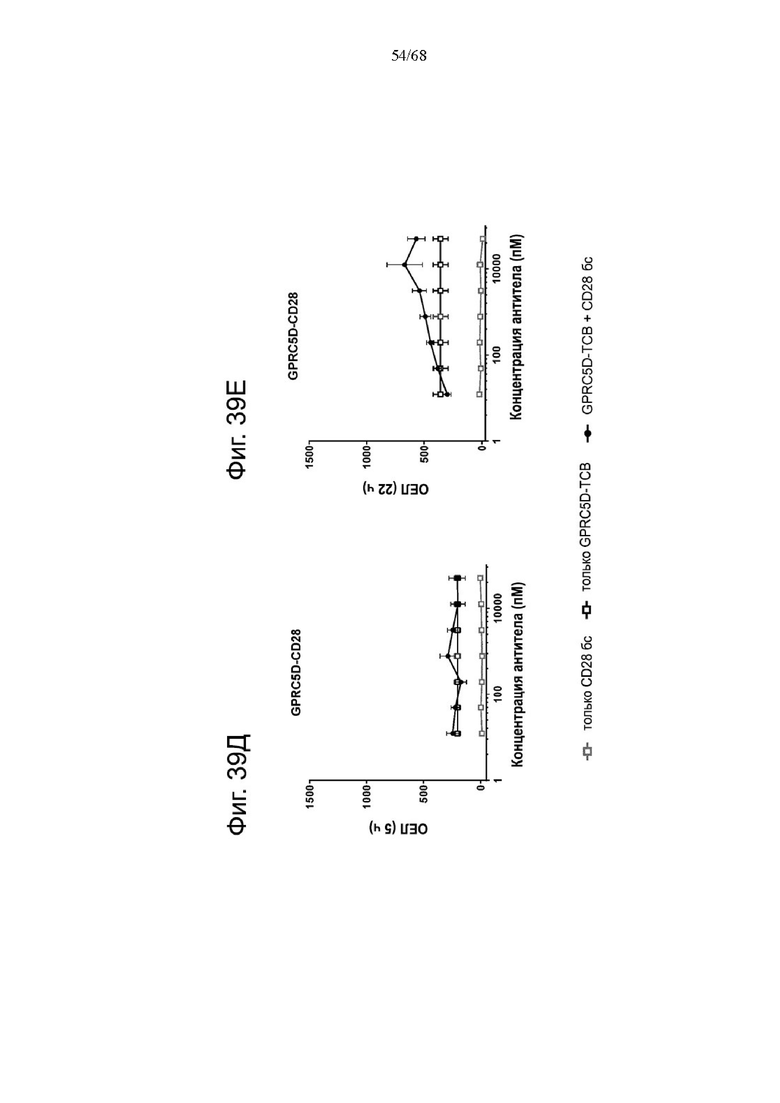
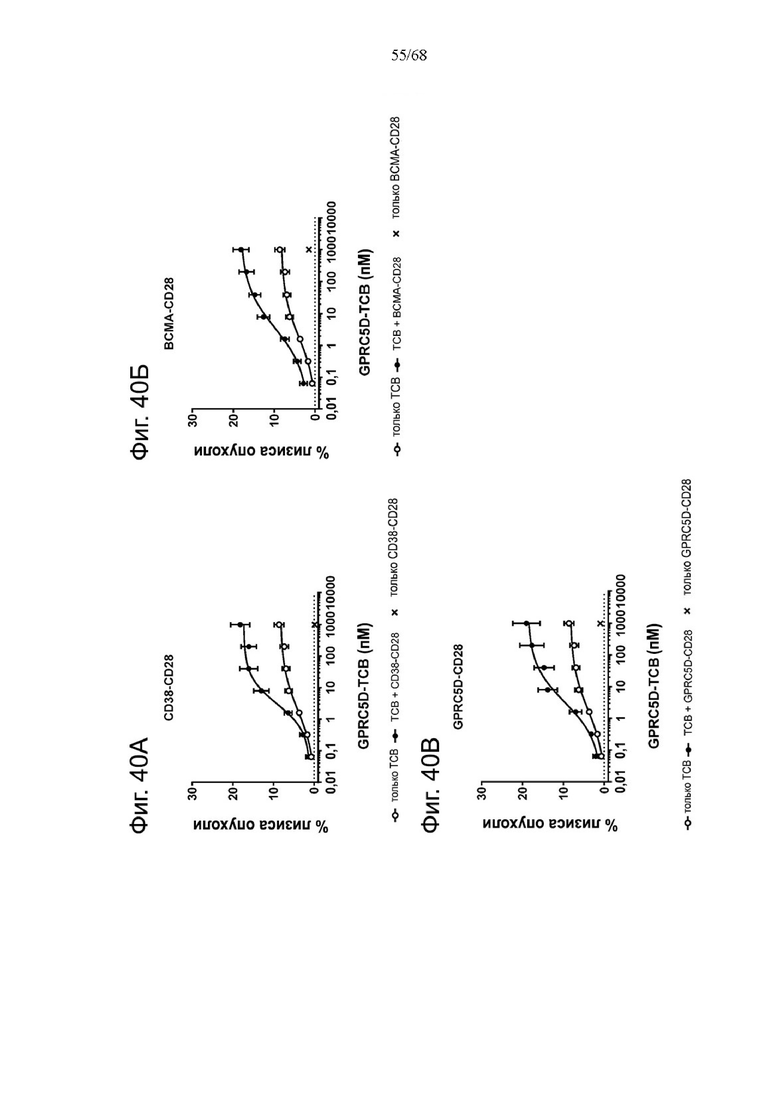
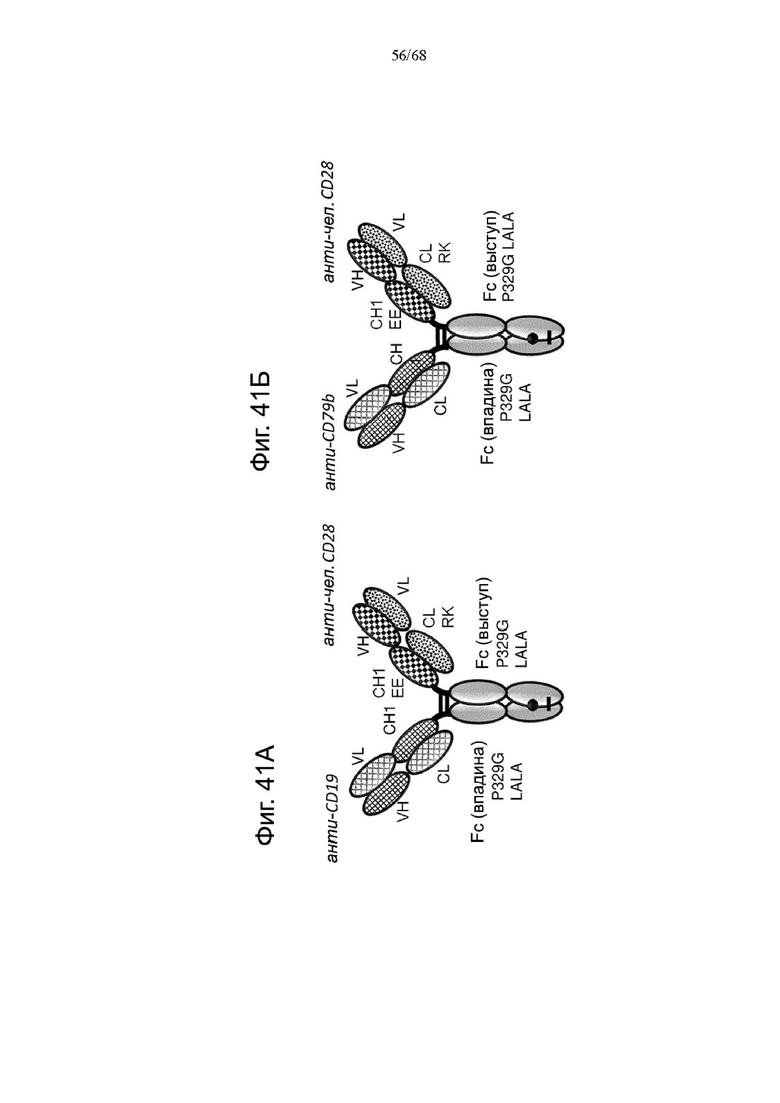
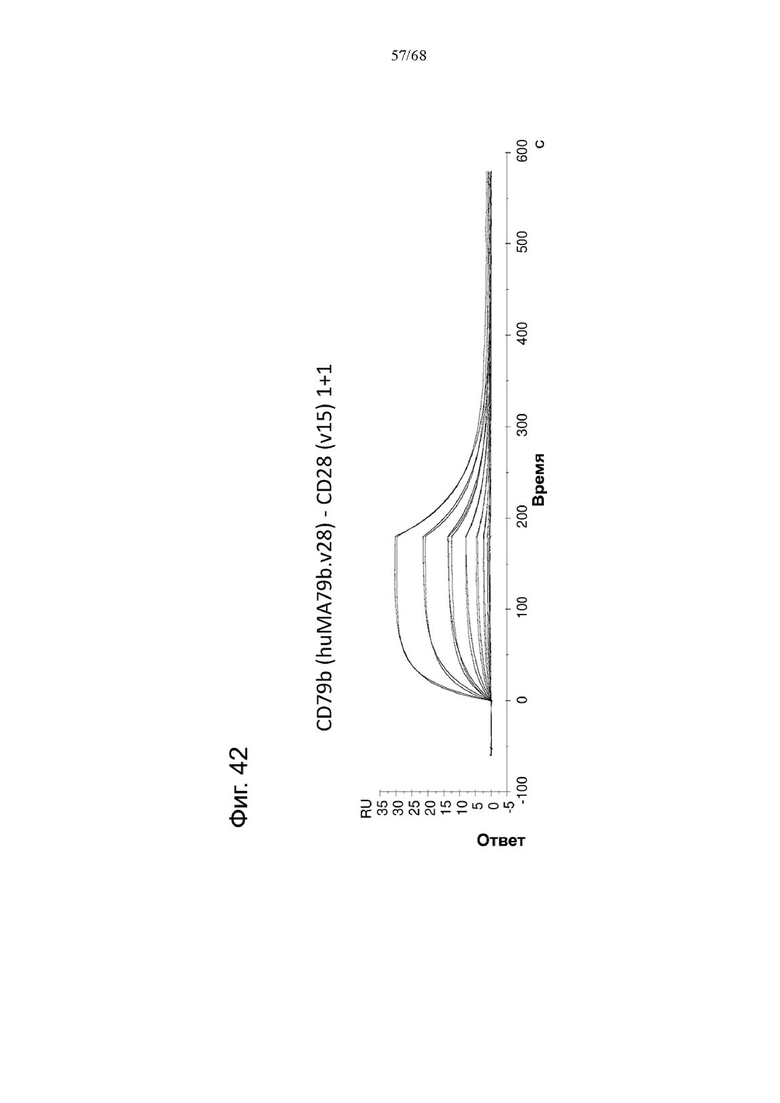
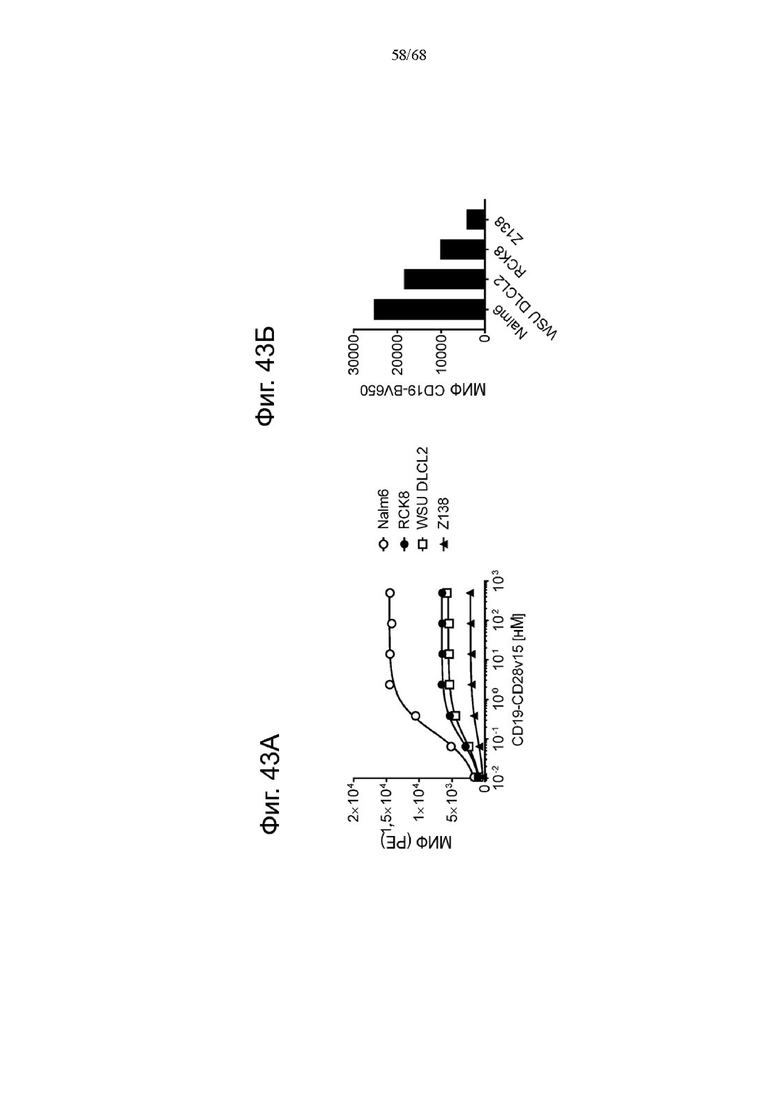
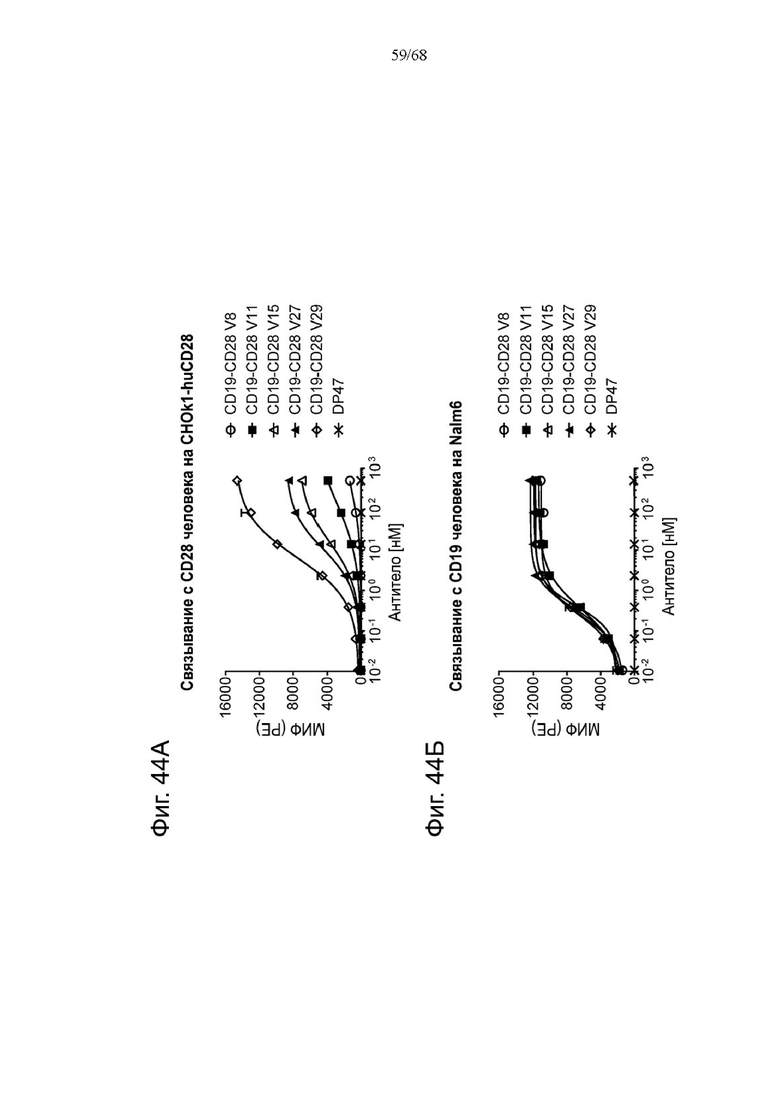
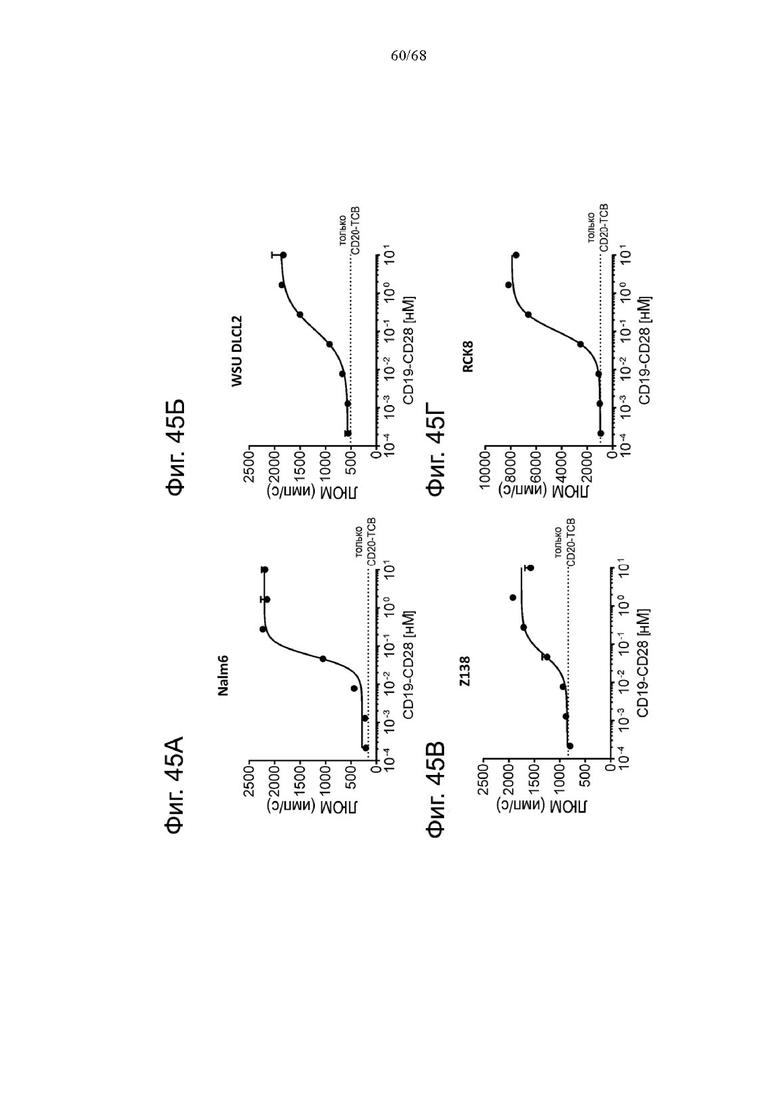
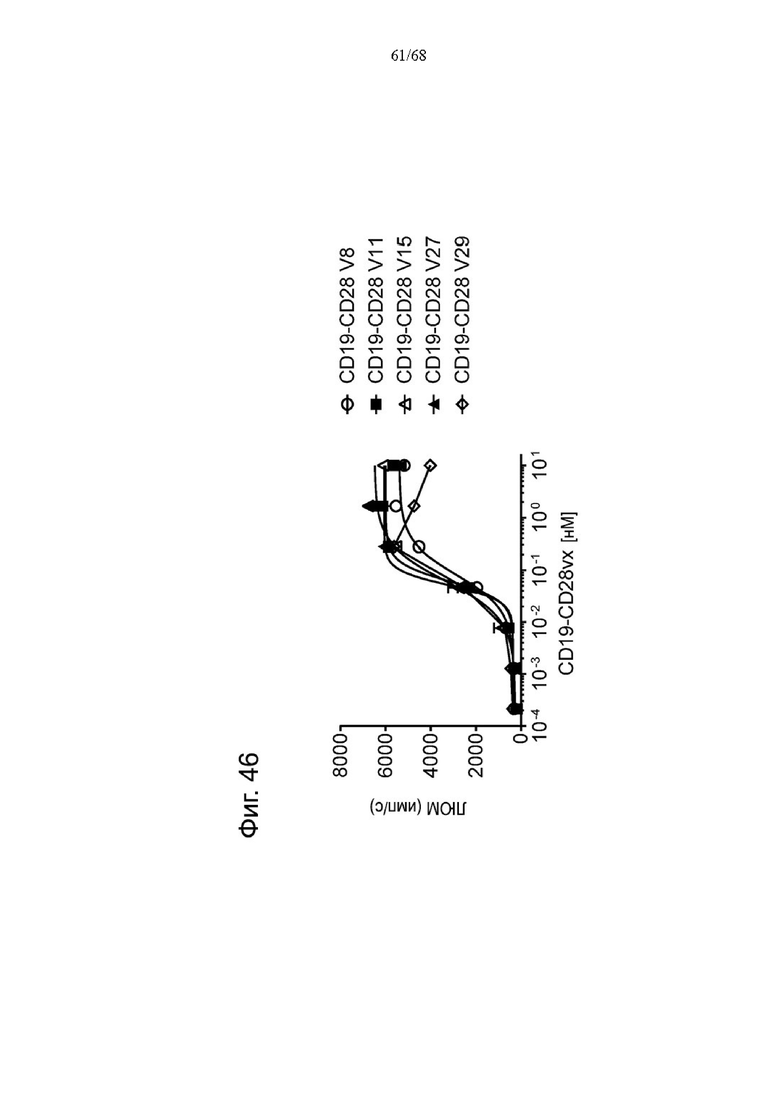
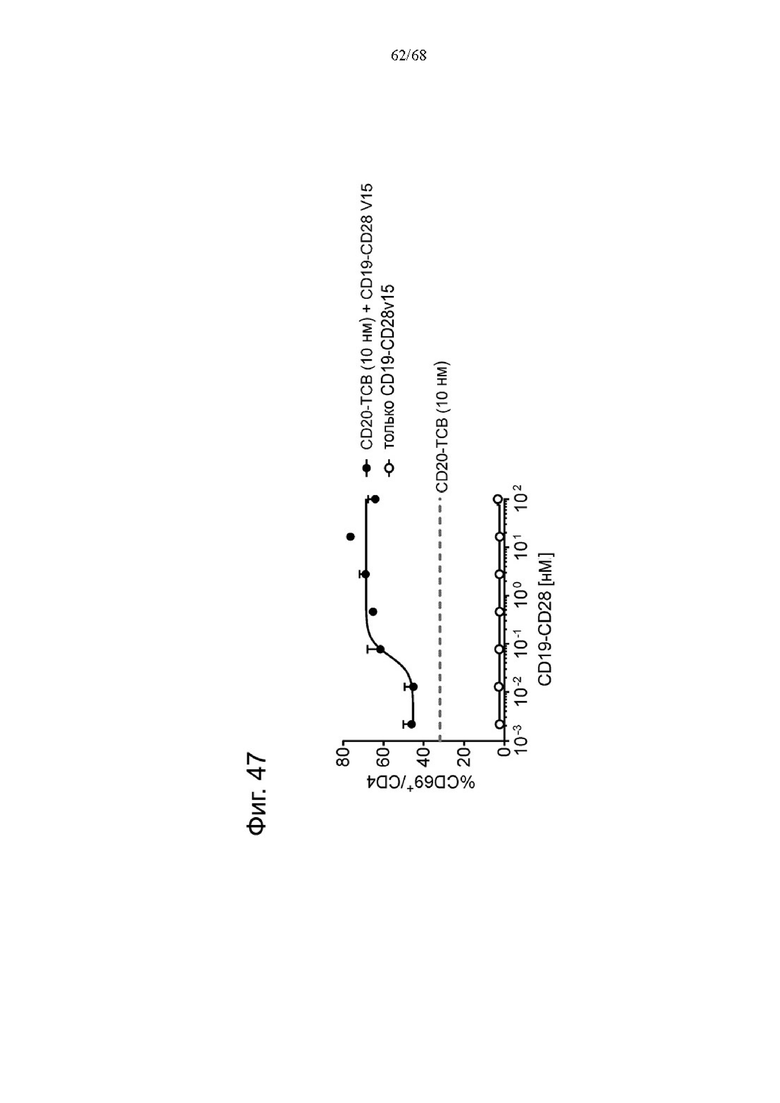
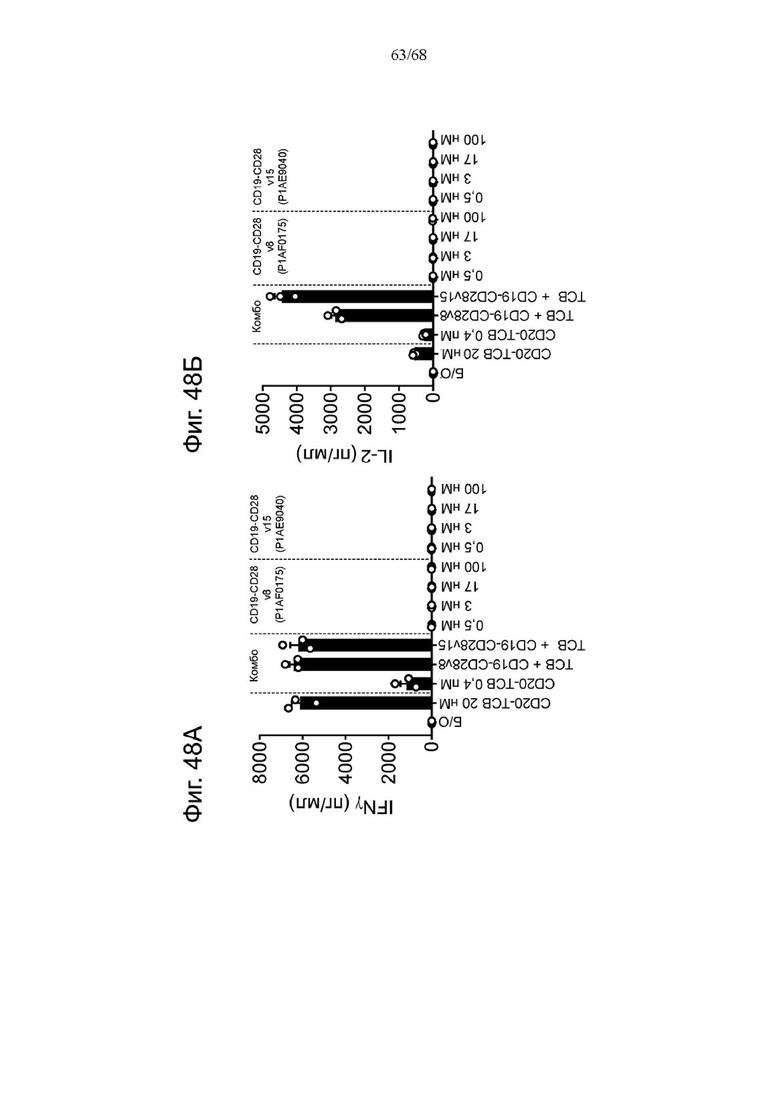
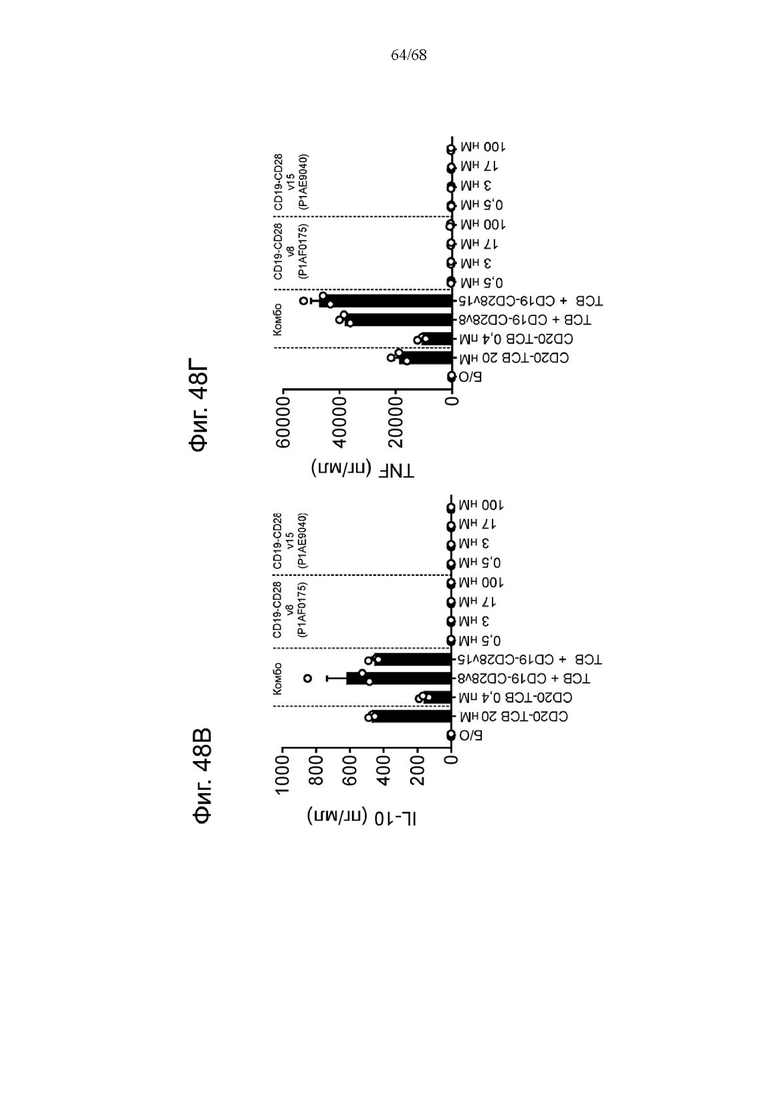
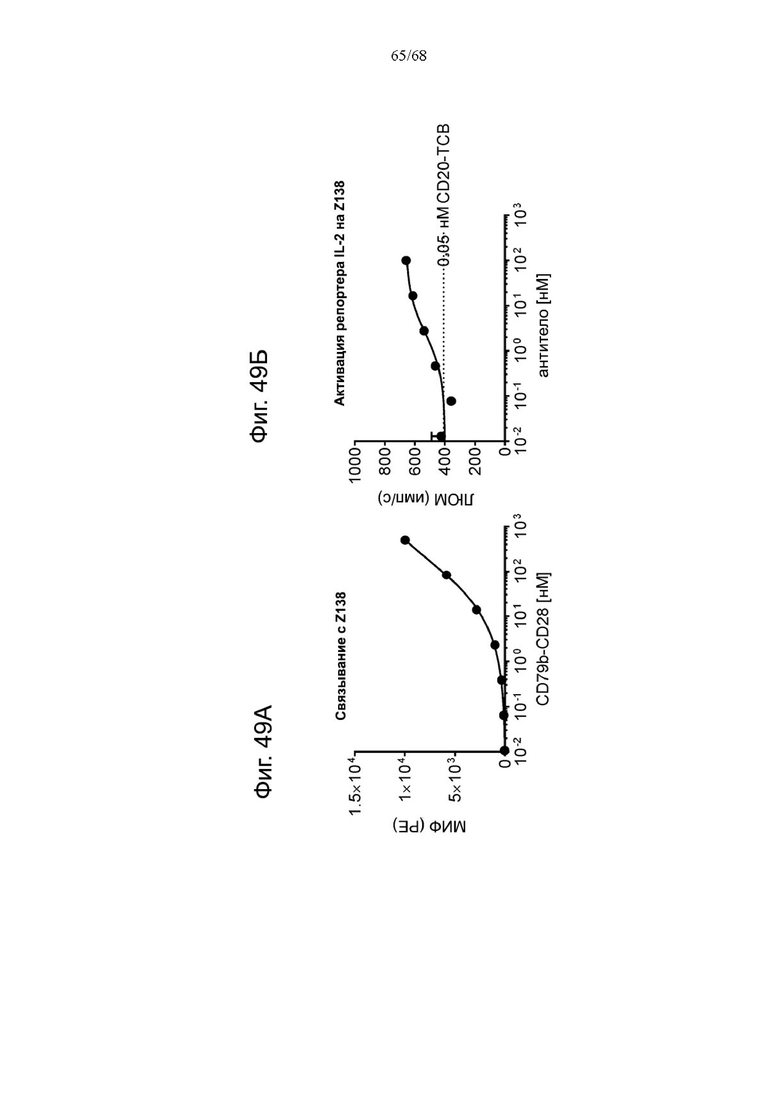
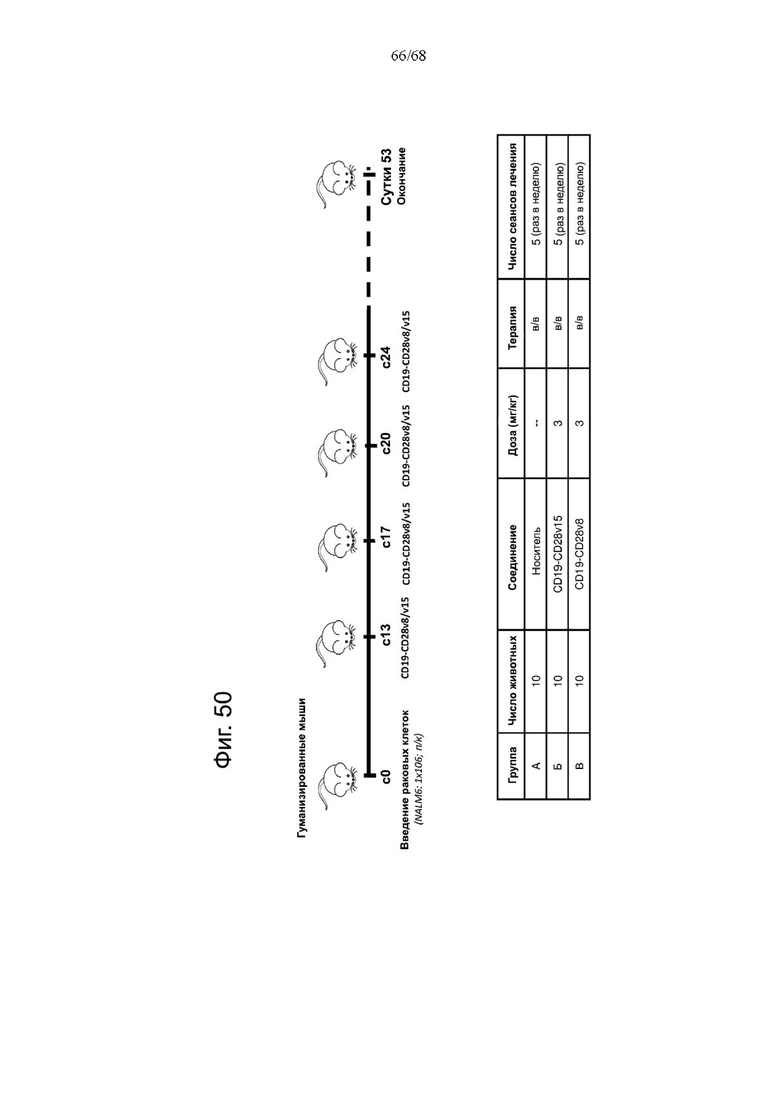
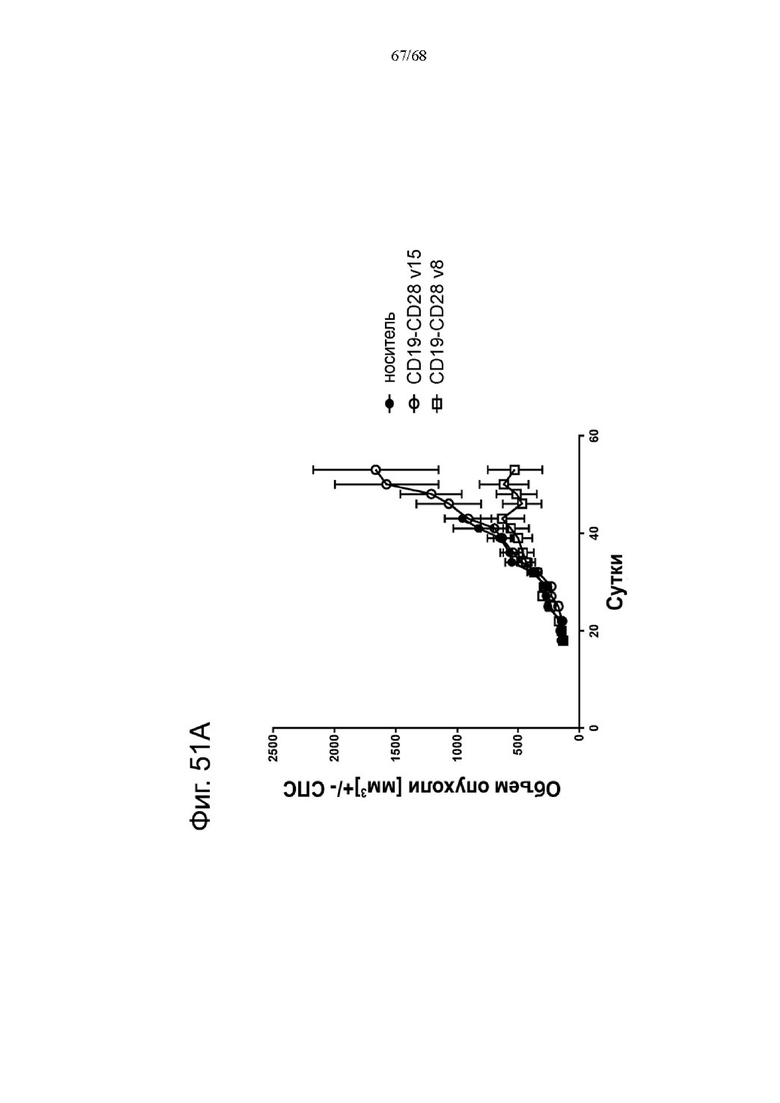
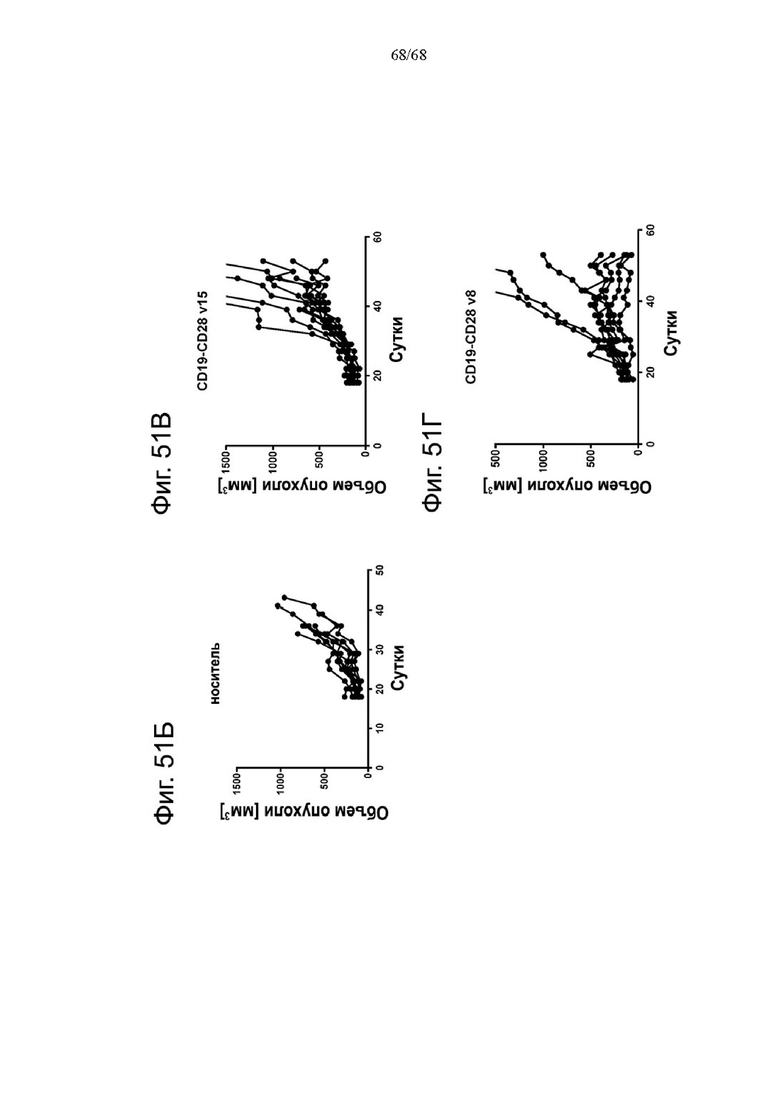
Изобретение относится к области биотехнологии. Предложена биспецифическая агонистическая CD28-антигенсвязывающая молекула, нацеленная на CD28 и CD19, характеризующаяся одновалентным связыванием с CD28. Также предложены полинуклеотид и вектор экспрессии, кодирующие указанную молекулу, и клетка-хозяин, содержащая их. Также изобретение относится к способу получения биспецифических молекул и способам их применения. Изобретение может быть использовано в терапии рака. 9 н. и 11 з.п. ф-лы, 157 ил., 46 табл., 22 пр.
1. Биспецифическая агонистическая CD28-антигенсвязывающая молекула, нацеленная на CD28 и CD19, характеризующаяся одновалентным связыванием с CD28, содержащая:
(а) один антигенсвязывающий домен, способный к специфическому связыванию с CD28, включающий
(i) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 47, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 54, или
(ii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 53, или
(iii) вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 42, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 27,
(б) один антигенсвязывающий домен, способный к специфическому связыванию с CD19, содержащий вариабельную область тяжелой цепи (VHCD19), содержащую аминокислотную последовательность SEQ ID NO: 412, и вариабельную область легкой цепи (VLCD19), содержащую аминокислотную последовательность SEQ ID NO: 413, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
2. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по п. 1, в которой Fc-домен принадлежит подклассу IgG1 человека и содержит аминокислотные мутации L234A, L235A и P329G (нумерация в соответствии с индексом EU по Kabat).
3. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по п.1 или 2, в которой Fab-фрагмент, способный специфически связываться с CD28, содержит вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 47, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 54.
4. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по п. 1 или 2, в которой Fab-фрагмент, способный специфически связываться с CD28, содержит вариабельную область тяжелой цепи (VHCD28), содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи (VLCD28), содержащую аминокислотную последовательность SEQ ID NO: 53.
5. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по п.1, содержащая первую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 122, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 114, вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 430, и вторую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 431.
6. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по п.1, содержащая аминокислотную последовательность SEQ ID NO: 121, первую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 116, вторую тяжелую цепь, содержащую аминокислотную последовательность SEQ ID NO: 430, и вторую легкую цепь, содержащую аминокислотную последовательность SEQ ID NO: 431.
7. Биспецифическая агонистическая CD28-антигенсвязывающая молекула по любому из пп. 1-6, содержащая:
(а) один Fab-фрагмент, способный к специфическому связыванию с CD28,
(б) один кросс-Fab-фрагмент, способный к специфическому связыванию с CD19, и
(в) Fc-домен, состоящий из первой и второй субъединиц, способных к стабильной ассоциации, содержащий одну или более аминокислотных замен, которые снижают аффинность связывания антигенсвязывающей молекулы с Fc-рецептором и/или эффекторную функцию.
8. Выделенный полинуклеотид, кодирующий биспецифическую агонистическую CD28-антигенсвязывающую молекулу по любому из пп. 1-7.
9. Вектор экспрессии, содержащий полинуклеотид по п. 8.
10. Клетка-хозяин для получения биспецифической агонистической CD28-антигенсвязывающей молекулы по любому из пп. 1-7, содержащая вектор по п. 9.
11. Способ получения биспецифической агонистической CD28-антигенсвязывающей молекулы, включающий этапы а) культивирования клетки-хозяина по п. 10 в условиях, подходящих для экспрессии биспецифической агонистической CD28-антигенсвязывающей молекулы, и б) выделения биспецифической агонистической CD28-антигенсвязывающей молекулы.
12. Применение фармацевтической композиции, содержащей биспецифическую агонистическую CD28-антигенсвязывающую молекулу по любому из пп. 1-7 и по меньшей мере один фармацевтически приемлемый эксципиент, для лечения рака.
13. Применение биспецифической агонистической CD28-антигенсвязывающей молекулы по любому из пп. 1-7 для повышения (а) активации T-клеток или (б) эффекторных функций T-клеток.
14. Применение биспецифической агонистической CD28-антигенсвязывающей молекулы по любому из пп. 1-7 для лечения рака.
15. Применение биспецифической агонистической CD28-антигенсвязывающей молекулы по любому из пп. 1-7 для лечения В-клеточного пролиферативного нарушения, выбранного из группы, состоящей из неходжкинской лимфомы (НХЛ), острого лимфолейкоза (ОЛЛ), хронического лимфоцитарного лейкоза, лейкоза (CLL), диффузной крупноклеточной B-клеточной лимфомы (DLBCL), фолликулярной лимфомы (FL), мантийно-клеточной лимфомы (MCL), лимфомы маргинальной зоны (MZL), множественной миеломы (MM) и лимфомы Ходжкина (HL).
16. Применение по п. 14 или 15, причем агонистическая CD28-антигенсвязывающая молекула предназначена для введения в комбинации с химиотерапевтическим агентом, лучевой терапией и/или другими агентами для применения в иммунотерапии рака.
17. Применение по п. 14 или 15, причем агонистическая CD28-антигенсвязывающая молекула предназначена для введения в комбинации с активирующим T-клетки биспецифическим антителом к CD3.
18. Применение по п.14 или 15, причем агонистическая CD28-антигенсвязывающая молекула предназначена для введения в комбинации с биспецифическим антителом анти-CD20/анти-CD3.
19. Применение по п. 14 или 15, причем агонистическая антигенсвязывающая молекула CD28 предназначена для введения в комбинации с биспецифическим антителом анти-CD20/анти-CD3, содержащим полипептидную последовательность SEQ ID NO: 455, полипептидную последовательность SEQ ID NO: 456, полипептидную последовательность SEQ ID NO: 457 и полипептидную последовательность SEQ ID NO: 458.
20. Применение по п. 14 или 15, причем агонистическая CD28-антигенсвязывающая молекула предназначена для введения в комбинации с антителом к PD-L1 или антителом к PD-1.
| US 2014242080 A1, 28.08.2014 | |||
| US 7635472 B2, 22.12.2009 | |||
| BOHLEN H | |||
| et al., Cytolysis of Leukemic B-Cells by T-Cells Activated via Two Bispecific Antibodies, Cancer Res, 1993, vol.53(18), pp.4310-4314 | |||
| REICHERT J.M., DHIMOLEA E., The future of antibodies as cancer drugs, Drug Discovery Today, 2012, Volume 17, Numbers 17/18, pp.954-963 |
Авторы
Даты
2023-11-22—Публикация
2019-12-19—Подача