Область техники, к которой относится изобретение
Настоящее изобретение относится к полипептидным комплексам, которые позволяют осуществлять лечение рака благодаря их способности приближать Т-клетки к раковым клеткам-мишеням и использованию цитотоксической активности Т-клеток в отношении раковых клеток-мишеней, к способам получения полипептидных комплексов и к терапевтическим агентам, которые содержат указанный полипептидный комплекс в качестве действующего вещества для индукции клеточной цитотоксичности. Настоящее изобретение относится также к фармацевтическим композициям, предназначенным для лечения или предупреждения различных видов рака, которые содержат в качестве действующего вещества указанный выше терапевтический агент для индукции клеточной цитотоксичности, и к терапевтическим способам, заключающимся в применении указанных фармацевтических композиций.
Предпосылки создания изобретения
К настоящему времени в качестве фармацевтических средств для лечения рака разработано множество терапевтических антител, обладающих очень высокой противоопухолевой активностью (непатентный документ 1). Известно, что эти терапевтические антитела проявляют свое противоопухолевое действие на раковые клетки посредством ингибирования сигналов, имеющих решающее значение для роста раковых клеток, индукции сигналов клеточной гибели, антитело-обусловленной клеточнозависимой цитотокичности (ADCC) или комплементзависимой цитотоксичности (CDC) (непатентный документ 2). ADCC представляет собой цитотоксичность, вызываемую эффекторными клетками, такими как NK-клетки и макрофаги, в отношении связанных с антителом раковых клеток-мишеней, когда Fc-область антитела связывается с Fc-рецептором на эффекторных клетках. При этом комплекс комплемента связывается с комплементсвязывающим сайтом в структуре антитела. CDC представляет собой цитотоксичность, которая имеет место в том случае, когда компонент системы комплемента в комплексе формирует пору в клеточной мембране связанной с антителом клетки, усиливающую приток воды или ионов в клетку. Хотя общепринятые терапевтические антитела обладают очень высокой активностью, до настоящего времени введение указанных антител приводило лишь к неудовлетворительным исходам терапевтического лечения. Таким образом, существует потребность в создании терапевтических антител с более высокой способностью к уничтожению раковых клеток.
Помимо указанных выше антител, которые приобретают ADCC в качестве их противоопухолевого механизма посредством рекрутмента NK-клеток или макрофагов в качестве эффекторных клеток, вызывающие рекрутмент Т-клеток антитела (TR-антитела), которые приобретают цитотоксичность в качестве их противоопухолевого механизма путем рекрутмента Т-клеток в качестве эффекторных клеток, известны с 1980-х годов (непатентный документы 3-5). TR-антитело представляет собой биспецифическое антитело, которое содержит антитело к любой из субъединиц, образующих комплекс Т-клеточного рецептора (TCR) на Т-клетках, в частности, антитело, которое связывается с эпсилон-цепью CD3, и антитело, которое связывается с антигеном на раковых клетках-мишенях. Т-клетка приближается к раковой клетке, когда TR-антитело одновременно связывается как с эпсилон-цепью CD3, так и с раковым антигеном, и это приводит к противоопухолевому действию в отношении раковой клетки благодаря цитотоксической активности Т-клетки.
В качестве TR-антитела известно также антитело, обозначенное как «трехфункциональное антитело» (непатентные документы 6 и 7). Трехфункциональное антитело представляет собой полное биспецифическое антитело IgG-типа, в котором одно плечо содержит Fab, связывающийся с раковым антигеном, а другое плечо содержит Fab, связывающийся с эпсилон-цепью CD3. Терапевтические действие в отношении злокачественных асцитов продемонстрировано при введении катумаксомаба, представляющего собой трехфункциональное антитело к ЕрСАМ, в перитонеальные полости пациентов со злокачественными асцитами, которые имеют позитивные по экспрессии ЕрСАМ раковые клетки. Применение катумаксомаба разрешено в странах Евросоюза для вышеуказанного лечения.
Кроме того, в настоящее время установлено, что TR-антитело, обозначенное как «биспецифическое, привлекающее Т-клетки антитело (BiTE)» обладает сильным противоопухолевым действием (не представляющие собой патент документы 8 и 9). BiTE представляет собой TR-антитело с молекулярной формой, в которой scFv антитела к раковому антигену сцеплен с scFv антитела к эпсилон-цепи CD3 через короткий полипептидный линкер. Известно, что BiTE обладают противоопухолевой активностью, превышающей активность различных известных TR-антител (непатентные документы 9 и 10). В частности, по сравнению с другими TR-антителами BiTE обладают противоопухолевой активностью даже при применении в существенно более низкой концентрации и при более низком соотношении эффекторных клеток/раковых клеток (ЕТ-соотношение). Продемонстрировано также, что их действие может с успехом проявляться без необходимости в предварительной активации эффекторных клеток с помощью IL-2, агонистического антитела к CD28 или др. Блинатумомаб (МТ103), который представляет собой BiTE к CD19, обладает более сильной цитотоксической активностью в отношении раковых клеток in vitro, чем ритуксан, который, как известно, обладает очень хорошим клиническим действием. Кроме того, чрезвычайно высокая активность блинатумомаба выявлена на фазе I и II проводимых в настоящее время клинических испытаний (непатентный документ 11).
На основе того факта, что катумаксомаб разрешен в качестве терапевтического агента, для которого в клинических условиях продемонстрировано лечебное действие, и что множество BiTE, включая блинатумомаб, обладают сильным противоопухолевым действием, высказано предположение о том, что TR-антитела, которые обеспечивают рекрутмент Т-клеток в качестве эффекторных клеток, могут обладать существенно более высоким потенциалом в качестве противоопухолевого агента по сравнению с общепринятыми антителами, механизм действия которых связан с ADCC.
Однако известно, что трехфункциональное антитело одновременно связывается как с Т-клеткой, так с такой клеткой как NK-клетка или макрофаг, независимым от ракового антигена образом, и в результате рецепторы, которые экспрессируются на клетках, являются перекрестносшитыми, и экспрессия различных цитокинов индуцируется независимым от ракового антигена образом. Предполагается, что системное введение трехфункционального антитела вызывает побочные действия типа «цитокинового шторма» в результате указанной индукции экспрессии цитокинов. Фактически, на фазе I клинического испытания было установлено, что максимальной переносимой дозой при системном введении катумаксомаба пациентам с немелкоклеточным раком легкого является очень низкая доза, составляющая 5 мкг/организм, и что введение более высокой дозы вызывает серьезные побочные действия (непатентный документ 12). При введении катумаксомаба в указанной низкой дозе его уровень в крови никогда не может достигать эффективного значения. Это означает, что ожидаемое противоопухолевое действие не может быть достигнуто при введении катумаксомаба в указанной низкой дозе.
При этом, в отличие от катумаксомаба, BiTE не содержит сайт связывания Fcγ-рецептора и поэтому у такого антитела отсутствует перекрестное сшивание с рецепторами, которые экспрессируются на Т-клетках и таких клетках, как NK-клетки и макрофаги, зависимым от ракового антигена образом. Так, было продемонстрировано, что BiTE не вызывает независимую от ракового антигена индукцию цитокинов, которая обнаружена при введении катумаксомаба. Однако, поскольку BiTE представляет собой модифицированную низкомолекулярную молекулу антитела без Fc-области, проблема заключается в том, что время его полужизни в крови после введения пациенту, существенно короче, чем в случае антител IgG-типа, которые обычно применяют в качестве терапевтических антител. Фактически, согласно опубликованным данным время полужизни в крови BiTE при его применении in vivo составляет примерно несколько часов (непатентные документы 13 и 14). При проведении клинических испытаний блинатумомаба его вводили путем непрерывной внутривенной инфузии с помощью мининасоса. Такой метод введения не только чрезвычайно неудобен для пациентов, но также имеет потенциальный риск медицинских осложнений, связанных с неисправностью устройства или т.п. Таким образом, нельзя считать, что указанный метод введения является желательным.
Документы, характеризующие известный уровень техники
[Непатентные документы]
[Непатентный документ 1]: Clin Cancer Res. 16 (1), 2010, сс. 11-20.
[Непатентный документ 2]: Drug Des Devel Ther 3, 2009, сс. 7-16.
[Непатентный документ 3]: Nature 314 (6012), 1985, сс. 628-631.
[Непатентный документ 4]: Int J Cancer 41 (4), 1988, сс. 609-615.
[Непатентный документ 5]: Proc Natl Acad Sci USA 83 (5), 1986, сс. 1453-1457.
[Непатентный документ 6]: Cancer Treat Rev. 36 (6), 2010, сс. 458-467.
[Непатентный документ 7]: Expert Opin Biol Ther 10 (8), 2010, сс. 1259-1269.
[Непатентный документ 8]: Proc Natl Acad Sci USA. 92 (15), 1995, cc. 7021-7025.
[Непатентный документ 9]: Drug Discov Today, 10 (18), 2005, cc. 1237-1244.
[Непатентный документ 10]: Trends Biotechnol 22 (5), 2004, cc. 238-244.
[Непатентный документ 11]: Science 321 (5891), 2008, cc. 974-977.
[Непатентный документ 12]: Cancer Immunol Immunother 56 (10), 2007, cc. 1637-1644.0
[Непатентный документ 13]: Cancer Immunol Immunother. 55 (5), 2006, cc. 503-514.
[Непатентный документ 14]: Cancer Immunol Immunother. 58 (1), 2009, cc. 95-109.
Краткое изложение сущности изобретения
Задачи, положенные в основу настоящего изобретения
Настоящее изобретение было создано с учетом вышеуказанных обстоятельств. В основу настоящего изобретения была положена задача создать пригодные для лечения рака полипептидные комплексы, которые обладают способностью приближать Т-клетки к раковой клетки-мишени и позволяют использовать цитотоксическую активность Т-клеток в отношении раковых клеток-мишеней, способы получения полипептидных комплексов и терапевтических агентов, которые содержат указанный полипептидный комплекс в качестве действующего вещества для индукции клеточной цитотоксичности. Другой задачей настоящего изобретения является разработка фармацевтических композиций, предназначенных для лечения или предупреждения различных видов рака, которые содержат в качестве действующего вещества указанный выше терапевтический агент для индукции цитотоксичности, и терапевтических способов, заключающихся в применении указанных фармацевтических композиций. Средства решения указанных задач
При создании изобретения были открыты новые полипептидные комплексы, которые сохраняют сильную противоопухолевую активность, присущую BiTE, и обладают длительным временем полужизни в крови, а также очень высокой безопасностью, что приводит к отсутствию индукции независимого от ракового антигена «цитокинового шторма» или т.п. При создании настоящего изобретения установлено также, что при замене антигенсвязывающих доменов полипептидных комплексов полипептидные комплексы могут повреждать различные клетки-мишени. Основываясь на указанных выше данных, при создании настоящего изобретения продемонстрировано, что полипептидные комплексы, предлагаемые в настоящем изобретении, повреждают раковые клетки. При создании настоящего изобретения установлено также, что путем регуляции ассоциации на поверхности раздела CH1/CL и интродукции модификаций типа «выступы-во-впадины» («выступ-впадина») (Knobs-into-Holes (KiH)) в полипептидные комплексы достигается более эффективная клеточная цитотоксичность. Кроме того, при создании настоящего изобретения продемонстрировано, что с использованием терапевтических агентов для индукции клеточной цитотоксичности, которые содержат в качестве действующего вещества полипептидный комплекс, предлагаемый в настоящем изобретении, можно лечить или предупреждать различные виды рака.
Более конкретно, в настоящем изобретении предложены:
[1] полипептидный комплекс, который содержит:
(1) антигенсвязывающий домен;
(2) домен, содержащий Fc-область, которая обладает пониженной активностью связывания с Fcγ-рецептором; и
(3) домен, связывающий комплекс Т-клеточного рецептора;
[2] полипептидный комплекс по п. [1], в котором домен, связывающий комплекс Т-клеточного рецептора, представляет собой домен, связывающий Т-клеточный рецептор;
[3] полипептидный комплекс по п. [1], в котором домен, связывающий комплекс Т-клеточного рецептора, представляет собой CD3-связывающий домен;
[4] полипептидный комплекс по одному из п.п. [1]-[3], в котором антигенсвязывающий домен представляет собой двухвалентный антигенсвязывающий домен;
[5] полипептидный комплекс по п. [4], в котором двухвалентный антигенсвязывающий домен представляет собой домен, имеющий структуру F(ab')2;
[6] полипептидный комплекс по п. [5], в котором два полипептида, образующие константную область тяжелой цепи домена, который имеет структуру F(ab')2, индивидуально сцеплены с любым из двух полипептидов, образующих Fc-домен;
[7] полипептидный комплекс по п. [6], в котором CD3-связывающий домен сцеплен с любым из двух или с обоими СН3, образующими Fc-домен;
[8] полипептидный комплекс по п. [7], в котором Fv-фрагмент тяжелой цепи, образующий CD3-связывающий домен, сцеплен с одним из СН3, образующих Fc-домен, и Fv-фрагмент легкой цепи, образующий CD3-связывающий домен, сцеплен с другим СН3, образующим Fc-домен;
[9] полипептидный комплекс по п. [8], в котором CH1-домен антитела сцеплен с Fv-фрагментом тяжелой цепи, образующим CD3-связывающий домен, и CL-домен антитела сцеплен с Fv-фрагментом легкой цепи;
[10] полипептидный комплекс по п. [6], в котором CD3-связывающий домен сцеплен с любым из двух или с обоими CL, образующими F(ab')2;
[11] полипептидный комплекс по п. [6], в котором CD3-связывающий домен сцеплен с любой из двух или с обеими VH, образующими F(ab')2;
[12] полипептидный комплекс по п. [6], в котором CD3-связывающий домен сцеплен с любой из двух или с обоими VL, образующими F(ab')2;
[13] полипептидный комплекс по одному из п.п. [1]-[12], в котором CD3-связывающий домен представляет собой Fv;
[14] полипептидный комплекс по одному из п.п. [1]-[7] и [10]-[12], в котором CD3-связывающий домен представляет собой Fab;
[15] полипептидный комплекс по одному из п.п. [1]-[7] и [10]- [12], в котором CD3-связывающий домен представляет собой scFv;
[16] полипептидный комплекс по одному из п.п. [1]-[15], в котором CD3-связывающий домен является одновалентным;
[17] полипептидный комплекс по одному из п.п. [1]-[3], в котором антигенсвязывающий домен представляет собой одновалентный scFv и одновалентный Fab;
[18] полипептидный комплекс по п. [17], в котором одновалентный scFv сцеплен с одним из полипептидов, образующих Fc-домен, через scFv, который образует CD3-связывающий домен; Fv-фрагмент тяжелой цепи одновалентного Fab сцеплен с одним из полипептидов, образующих Fc-домен, через СН1-домен; и Fv-фрагмент легкой цепи Fab сцеплен с CL-доменом;
[19] полипептидный комплекс по одному из п.п. [1]-[3], в котором антигенсвязывающий домен представляет собой двухвалентный scFv;
[20] полипептидный комплекс по п. [19], в котором один одновалентный scFv сцеплен с одним из полипептидов, образующих Fc-домен, через Fv-фрагмент тяжелой цепи, образующий CD3-связывающий домен, и другой одновалентный scFv сцеплен с другим полипептидом, образующим Fc-домен, через Fv-фрагмент легкой цепи, образующий CD3-связывающий домен;
[21] полипептидный комплекс по п. [19], в котором один одновалентный scFv сцеплен с одним из полипептидов, образующих Fc-домен, через scFv, образующий CD3-связывающий домен, и другой одновалентный scFv сцеплен с другим полипептидом, образующим Fc-домен;
[22] полипептидный комплекс по одному из п.п. [1]-[3], в котором антигенсвязывающий домен и домен, связывающий комплекс Т-клеточного рецептора, каждый представляет собой одновалентный Fab;
[23] полипептидный комплекс по п. [22], в котором Fv-фрагмент тяжелой цепи одновалентного Fab, образующего антигенсвязывающий домен, сцеплен с одним из полипептидов, образующих Fc-домен, через CH1-домен и Fv-фрагмент легкой цепи Fab сцеплен с CL-доменом; и Fv-фрагмент тяжелой цепи Fab, образующего домен, связывающий Т-клеточный рецептор, сцеплен с другим полипептидом, образующим Fc-домен, через CH1-домен и Fv-фрагмент легкой цепи Fab сцеплен с CL-доменом;
[24] полипептидный комплекс по п. [22], в котором Fv-фрагмент одновалентного Fab, образующего антигенсвязывающий домен, сцеплен с одним из полипептидов, образующих Fc-домен, через CH1-домен и Fv-фрагмент легкой цепи Fab сцеплен с CL-доменом; и Fv-фрагмент легкой цепи Fab, образующего домен, связывающий Т-клеточный рецептор, сцеплен с другим полипептидом, образующим Fc-домен, через CH1-домен и Fv-фрагмент тяжелой цепи Fab сцеплен с CL-доменом;
[25] полипептидный комплекс по п. [22], в котором Fv-фрагмент тяжелой цепи одновалентного Fab, образующего антигенсвязывающий домен, сцеплен с одним из полипептидов, образующих Fc-домен, через CH1-домен и Fv-фрагмент легкой цепи Fab сцеплен с CL-доменом; и Fv-фрагмент тяжелой цепи Fab, образующего домен, связывающий Т-клеточный рецептор, сцеплен с другим полипептидом, образующим Fc-домен, через CL-домен и Fv-фрагмент легкой цепи Fab сцеплен с СН1-доменом;
[26] полипептидный комплекс по п. [22], в котором Fv-фрагмент тяжелой цепи одновалентного Fab, образующего домен, связывающий Т-клеточный рецептор, сцеплен с одним из полипептидов, образующих Fc-домен, через CH1-домен и Fv-фрагмент легкой цепи Fab сцеплен с CL-доменом; и Fv-фрагмент легкой цепи Fab, образующего антигенсвязывающий домен, сцеплен с другим полипептидом, образующим Fc-домен, через CH1-домен и Fv-фрагмент тяжелой цепи Fab сцеплен с CL-доменом;
[27] полипептидный комплекс по п. [22], в котором Fv-фрагмент тяжелой цепи одновалентного Fab, образующего домен, связывающий Т-клеточный рецептор, сцеплен с одним из полипептидов, образующих Fc-домен, через CH1-домен; и Fv-фрагмент легкой цепи Fab сцеплен с CL-доменом; и Fv-фрагмент тяжелой цепи Fab, образующего антигенсвязывающий домен, сцеплен с другим полипептидом, образующим Fc-домен, через CL-домен, и Fv-фрагмент легкой цепи Fab сцеплен с СН1-доменом;
[28] полипептидный комплекс по п. [22] который содержит:
(1) антигенсвязывающий домен, в котором Fv-фрагмент тяжелой цепи одновалентной структуры Fab, который связывается с антигеном, сцеплен через CH1-домен с одним из полипептидов, образующих Fc-домен, и Fv-фрагмент легкой цепи структуры Fab сцеплен с CL-доменом; и
(2) домен, связывающий комплекс Т-клеточного рецептора, в котором Fv-фрагмент тяжелой цепи одновалентной структуры Fab, который связывается с комплексом Т-клеточного рецептора, сцеплен через СН1 с другим полипептидом, образующим Fc-домен, и Fv-фрагмент легкой цепи структуры Fab сцеплен с CL-доменом;
при этом электрические заряды СН1- и CL-доменов контролируют таким образом, чтобы Fv-фрагмент тяжелой цепи антигенсвязывающего домена соединялся с Fv-фрагментом легкой цепи антигенсвязывающего домена или Fv-фрагмент тяжелой цепи домена, связывающего Т-клеточный рецептор, соединялся с Fv-фрагментом легкой цепи домена, связывающего Т-клеточный рецептор;
[29] полипептидный комплекс по п. [28], в котором аминокислотный остаток в CH1-домене, сцепленный с Fv-фрагментом тяжелой цепи домена, связывающего комплекс Т-клеточного рецептора, имеет такой же тип электрического заряда, что аминокислотный остаток в CL-домене, сцепленный с Fv-фрагментом антигенсвязывающего домена;
[30] полипептидный комплекс по п. [28], в котором аминокислотный остаток в CH1-домене, сцепленный с Fv-фрагментом тяжелой цепи антигенсвязывающего домена, имеет такой же тип электрического заряда, что аминокислотный остаток в CL-домене, сцепленный с Fv-фрагментом легкой цепи домена, связывающего комплекс Т-клеточного рецептора;
[31] полипептидный комплекс по [28], в котором аминокислотный остаток в CH1-домене, сцепленный с Fv-фрагментом тяжелой цепи домена, связывающего комплекс Т-клеточного рецептора, имеет такой же тип электрического заряда, что аминокислота в CL-домене, сцепленная с Fv-фрагментом легкой цепи антигенсвязывающего домена, и аминокислотный остаток в CH1-домене, сцепленный с Fv-фрагментом тяжелой цепи антигенсвязывающего домена, имеет такой же тип электрического заряда, что аминокислота в CL-домене, сцепленная с Fv-фрагментом легкой цепи домена, связывающего комплекс Т-клеточного рецептора;
[32] полипептидный комплекс по п. [29] или п. [31], в котором аминокислотный остаток в СН1-домене, сцепленный с Fv-фрагментом тяжелой цепи домена, связывающего комплекс Т-клеточного рецептора, имеет электрический заряд, противоположный заряду аминокислотного остатка в CL-домене, сцепленного с Fv-фрагментом легкой цепи связывающего Т-клеточный рецептор домена;
[33] полипептидный комплекс по п. [30] или п. [31], в котором аминокислотный остаток в СН1-домене, сцепленный с Fv-фрагментом тяжелой цепи антигенсвязывающего домена, имеет электрический заряд, противоположный заряду аминокислотного остатка в CL-домене, сцепленного с Fv-фрагментом легкой цепи антигенсвязывающего домена;
[34] полипептидный комплекс по одному из п.п. [22]-[33], в котором домен, связывающий комплекс Т-клеточного рецептора, представляет собой домен, связывающий Т-клеточный рецептор;
[35] полипептидный комплекс по п. [34], в котором домен, связывающий Т-клеточный рецептор, представляет собой CD3-связывающий домен;
[36] полипептидный комплекс по п. [32] или п. [33], в котором аминокислотные остатки в СН1- и CL-доменах представляют собой одну, две или большее количество комбинаций аминокислотных остатков, выбранных из группы, включающей комбинации:
(а) аминокислотного остатка в положении 147 (EU-нумерация) в СН1-домене и аминокислотного остатка в положении 180 (EU-нумерация) в CL-домене;
(б) аминокислотного остатка в положении 147 (EU-нумерация) в СН1-домене и аминокислотного остатка в положении 131 (EU-нумерация) в CL-домене;
(в) аминокислотного остатка в положении 147 (EU-нумерация) в СН1-домене и аминокислотного остатка в положении 164 (EU-нумерация) в CL-домене;
(г) аминокислотного остатка в положении 147 (EU-нумерация) в CH1-домене и аминокислотного остатка в положении 138 (EU-нумерация) в CL-домене;
(д) аминокислотного остатка в положении 147 (EU-нумерация) в СН1-домене и аминокислотного остатка в положении 123 (EU-нумерация) в CL-домене; и
(е) аминокислотного остатка в положении 175 (EU-нумерация) в CH1-домене и аминокислотного остатка в положении 160 (EU-нумерация) в CL-домене;
и в котором аминокислотный остаток в CH1-домене имеет электрический заряд, противоположный заряду аминокислотного остатка в CL-домене;
[37] полипептидный комплекс по п. [36], в котором аминокислотные остатки выбраны из группы, дополнительно включающей комбинацию аминокислотных остатков:
(ж) аминокислотного остатка в положении 213 (EU-нумерация) в СН1-домене и аминокислотного остатка в положении 123 (EU-нумерация) в CL-домене;
[38] полипептидный комплекс по п. [36] или п. [37], в котором аминокислотный остаток, имеющий противоположный электрический заряд, выбран из аминокислотного остатка, входящего в любую из групп:
(X) глутаминовая кислота (Е) и аспарагиновая кислота (D); или
(Y) лизин (K), аргинин (R) и гистидин (Н);
[39] полипептидный комплекс по одному из п.п. [36]-[38], в котором аминокислотные остатки, имеющие противоположный электрический заряд, представляют собой:
Lys в положении 175 (EU-нумерация) в CH1-домене и Glu в положениях 131, 160 и 180 (EU-нумерация) в CL-домене;
[40] полипептидный комплекс по одному из п.п. [36]-[38], в котором аминокислотные остатки, имеющие противоположный электрический заряд, представляют собой:
Glu в положениях 147 и 175 (EU-нумерация) в CH1-домене и Lys в положениях 131, 160 и 180 (EU-нумерация) в CL-домене;
[41] полипептидный комплекс по п. [40], в котором аминокислотный остаток в положении 213 (EU-нумерация) в СН1-домене представляет собой Glu и аминокислотный остаток в положении 123 (EU-нумерация) в CL-домене представляет собой Lys;
[42] полипептидный комплекс по одному из п.п. [1]-[41], в котором Fc-домен характеризуется пониженной активностью связывания с Fcγ-рецептором в отношении FcγI, FcγIIA, FcγIIB, FcγIIIA и/или FcγIIIB;
[43] полипептидный комплекс по одному из п.п. [1]-[42], в котором Fc-домен представляет собой любой из Fc-доменов, имеющих SEQ ID NO: 23, 24, 25 и 26, в котором аминокислота(ы), образующая(ие) Fc-домен, изменена(ы) в результате мутации;
[44] полипептидный комплекс по п. [43], в котором Fc-домен содержит любую из указанных ниже аминокислот:
аминокислотную последовательность, простирающуюся от положения 118 до положения 260 (EU-нумерация), которая представляет собой последовательность SEQ ID NO: 24; или
аминокислотную последовательность, простирающуюся от положения 261 до положения 447 (EU-нумерация), которая представляет собой последовательность SEQ ID NO: 26;
[45] полипептидный комплекс по п. [43], в котором аминокислоты, образующие Fc-домен, содержат мутацию в любом из следующих положений: 220, 226, 229, 231, 232, 233, 234, 235, 236, 237, 238, 239, 240, 264, 265, 266, 267, 269, 270, 295, 296, 297, 298, 299, 300, 325, 327, 328, 329, 330, 331 и 332 (EU-нумерация);
[46] полипептидный комплекс по п. [45], в котором Fc-домен содержит мутацию в аминокислотах SEQ ID NO: 23, образующих Fc-домен;
[47] полипептидный комплекс по п. [46], в котором Fc-домен представляет собой Fc-домен, содержащий аминокислотную замену в положении 233, 234, 235, 236, 237, 327, 330 или 331 (EU-нумерация) на аминокислоту, присутствующую в соответствующем положении (EU-нумерация) в соответствующем IgG2 или IgG4;
[48] полипептидный комплекс по п. [46], в котором Fc-домен содержит аминокислотную мутацию в положении 234, 235 или 297 (EU-нумерация);
[49] полипептидный комплекс по п. [48], в котором аминокислота(ы) в положении 234, 235 и/или 297 заменена(ы) на аланин;
[50] полипептидный комплекс по одному из п.п. [43]-[49], в котором последовательности двух полипептидов, образующих Fc-домен, отличаются друг от друга;
[51] полипептидный комплекс по одному из п.п. [1]-[50], в котором аминокислота в положении 349 заменена на цистеин и аминокислота в положении 366 (EU-нумерация) заменена на триптофан в последовательности аминокислотных остатков одного из двух полипептидов, образующих Fc-домен; и в котором аминокислота в положении 356 заменена на цистеин, аминокислота в положении 366 заменена на серии, аминокислота в положении 368 заменена на аланин и аминокислота в положении 407 (EU-нумерация) заменена на валин в последовательности аминокислотных остатков другого полипептида;
[52] полипептидный комплекс по одному из п.п. [1]-[50], в котором аминокислота в положении 356 (EU-нумерация) заменена на лизин в последовательности аминокислотных остатков одного из двух полипептидов, образующих Fc-домен; аминокислота в положении 439 (EU-нумерация) заменена на глутаминовую кислоту в другом полипептиде; и аминокислота в положении 435 (EU-нумерация) заменена на аргинин в последовательности аминокислотных остатков любого из двух полипептидов;
[53] полипептидный комплекс по п. [51] или п. [52], в котором последовательность GK удалена в результате делеции из карбоксильных концов двух полипептидов, образующих Fc-домен;
[54] полипептидный комплекс по одному из п.п. [1]-[53], в котором антигенсвязывающие домены связываются с одним и тем же эпитопом;
[55] полипептидный комплекс по [54], где указанный эпитоп присутствует в белке, содержащем аминокислотную последовательность SEQ ID NO: 2;
[56] полипептидный комплекс по [54], где указанный эпитоп присутствует в белке, содержащем аминокислотную последовательность SEQ ID NO: 4;
[57] полипептидный комплекс по одному из п.п. [1] to [53], в котором каждый из антигенсвязывающих доменов связывается с различным (другим) эпитопом;
[58] полипептидный комплекс по п. [57], где указанный другой эпитоп присутствует в белке, содержащем аминокислотную последовательность SEQ ID NO: 2;
[59] полипептидный комплекс по п. [57], где другой эпитоп присутствует в белке, содержащем аминокислотную последовательность SEQ ID NO: 4;
[60] полинуклеотид, кодирующий полипептидный комплекс по одному из п.п. [1]-[59];
[61] вектор, содержащий полинуклеотид по п. [60]; [62] клетка, содержащая вектор по п. [61];
[63] способ получения полипептидного комплекса, заключающийся в том, что культивируют клетку по п. [62] и выделяют полипептидный комплекс из супернатанта культуры;
[64] терапевтический агент для индукции клеточной цитотоксичности, который содержит в качестве действующего вещества полипептидный комплекс по одному из п.п. [1]-[59];
[65] терапевтический агент по п. [64], где терапевтический агент для индукции клеточной цитотоксичности представляет собой терапевтический агент, применяемый при раке;
[66] терапевтический агент по п. [65], где рак представляет собой рак печени или рак легкого;
[67] способ лечения или предупреждения рака, заключающийся в том, что полипептидный комплекс по одному из п.п. [1]-[59] вводят пациенту, который нуждается в этом; и
[68] терапевтический или профилактический способ по п. [67], в котором рак представляет собой рак печени или рак легкого.
Настоящее изобретение относится также у наборам, предназначенным для применения в способе, предлагаемом в настоящем изобретении, которые содержат полипептидный комплекс, предлагаемый в настоящем изобретении, или полипептидный комплекс, полученный с помощью способа, предлагаемого в настоящем изобретении. Настоящее изобретение относится также к применению полипептидного комплекса, предлагаемого в настоящем изобретении, или полипептидного комплекса, полученного с помощью способа, предлагаемого в настоящем изобретении, для приготовления терапевтического агента для индукции клеточной цитотоксичности. Настоящее изобретение относится также к полипептидным комплексам, предлагаемым в настоящем изобретении, или полипептидным комплексам, полученным с помощью способов, предлагаемых в изобретении, для применения в способе, предлагаемом в настоящем изобретении.
Результаты изобретения
В настоящем изобретении предложены новые полипептидные комплексы, которые сохраняют сильную противоопухолевую активность BiTE и обладают длительным временем полужизни в крови, а также очень высокой безопасностью, что приводит к отсутствию индукции независимого от ракового антигена «цитокинового шторма» или т.п.Когда изменяют антигенсвязывающий домен полипептидного комплекса, предлагаемого в настоящем изобретении, то терапевтические агенты, которые содержат полипептидный комплекс в качестве действующего вещества для индукции клеточной цитотоксичности, могут направленно воздействовать и повреждать различные клетки, включая раковые клетки. Таким образом, можно лечить или предупреждать различные виды рака. Это обеспечивает требуемое лечения, которое является высоко безопасным и удобным и снижает физическую нагрузку на пациентов.
Краткое описание чертежей
На чертежах показано:
на фиг.1 - график, на котором представлено сравнение цитотоксической активности следующих антител к GPC3 (антиген, принадлежащий к семейству GPI-заякоренных рецепторов): GPC3 ERY1 (GPC3 BiTE), GPC3 ERY2 и антитела к GPC3 IgG-типа. Закрашенным квадратом 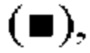 закрашенным треугольником
закрашенным треугольником  и незакрашенным квадратом
и незакрашенным квадратом 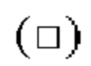 обозначена цитотоксическая активность GPC3 ERY1 (GPC3 BiTE), GPC3 ERY2 и антитела к GPC3 IgG-типа соответственно;
обозначена цитотоксическая активность GPC3 ERY1 (GPC3 BiTE), GPC3 ERY2 и антитела к GPC3 IgG-типа соответственно;
на фиг.2 график, на котором представлено сравнение цитотоксической активности GPC3 BiTE и GPC3 ERY5. Закрашенным квадратом 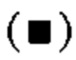 и незакрашенным кружком
и незакрашенным кружком 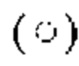 обозначена цитотоксическая активность GPC3 BiTE и GPC3 ERY5 соответственно;
обозначена цитотоксическая активность GPC3 BiTE и GPC3 ERY5 соответственно;
на фиг.3 - график, на котором представлено сравнение цитотоксической активности GPC3 BiTE и GPC3 ERY6. Закрашенным квадратом 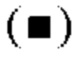 и закрашенным треугольником
и закрашенным треугольником  обозначена цитотоксическая активность GPC3 BiTE и GPC3 ERY6 соответственно;
обозначена цитотоксическая активность GPC3 BiTE и GPC3 ERY6 соответственно;
на фиг.4 график, на котором представлено сравнение цитотоксической активности GPC3 BiTE и GPC3 ERY7. Закрашенным квадратом 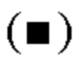 и закрашенным ромбом
и закрашенным ромбом  обозначена цитотоксическая активность GPC3 BiTE и GPC3 ERY7 соответственно;
обозначена цитотоксическая активность GPC3 BiTE и GPC3 ERY7 соответственно;
на фиг.5 - график, на котором представлено сравнение цитотоксической активности GPC3 BiTE, GPC3 ERY8-2, GPC3 ERY9-1 и GPC3 ERY10-1. Закрашенным квадратом 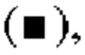 закрашенным треугольником
закрашенным треугольником 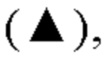 незакрашенной окружностью (○) и незакрашенным квадратом (□) обозначена цитотоксическая активность GPC3 BiTE, GPC3 ERY8-2, GPC3 ERY9-1 и GPC3 ERY10-1 соответственно;
незакрашенной окружностью (○) и незакрашенным квадратом (□) обозначена цитотоксическая активность GPC3 BiTE, GPC3 ERY8-2, GPC3 ERY9-1 и GPC3 ERY10-1 соответственно;
на фиг.6 график, демонстрирующий противоопухолевое действие in vivo GPC3 ERY8-2 на предварительно смешанной (pre-mix) модели РС-10. Незакрашенным квадратом (□) и закрашенным ромбом 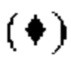 обозначены изменения объема опухолей в группе, для обработки которой применяли GPC3 ERY7, и контрольной (обработка ЗФР) группе соответственно;
обозначены изменения объема опухолей в группе, для обработки которой применяли GPC3 ERY7, и контрольной (обработка ЗФР) группе соответственно;
на фиг.7 - график, демонстрирующий противоопухолевое действие in vivo GPC3 ERY10-1 на предварительно смешанной (pre-mix) модели РС-10. Незакрашенным квадратом (□) и закрашенным ромбом 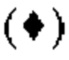 обозначены изменения объема опухолей в группе, для обработки которой применяли GPC3 ERY10-1, и контрольной (обработка ЗФР) группе соответственно;
обозначены изменения объема опухолей в группе, для обработки которой применяли GPC3 ERY10-1, и контрольной (обработка ЗФР) группе соответственно;
на фиг.8 - график, демонстрирующий противоопухолевое действие in vivo GPC3 ERY10-1 при моделировании на РС-10 Т-клеточного переноса. Незакрашенным квадратом (□) и закрашенным ромбом 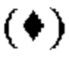 обозначены изменения объема опухолей в группе, для обработки которой применяли GPC3 ERY10-1, и контрольной (обработка ЗФР) группе соответственно;
обозначены изменения объема опухолей в группе, для обработки которой применяли GPC3 ERY10-1, и контрольной (обработка ЗФР) группе соответственно;
на фиг.9 график, на котором представлена зависимость от времени концентраций в плазме GPC3 ERY9-1 и GPC3 ERY10-1, определенных с использованием экспрессирующих GPC3 клеток Ba/F3. Закрашенным ромбом  и незакрашенным квадратом (□) обозначена зависимость от времени концентрации в плазме GPC3 ERY9-1 и GPC3 ERY10-1 соответственно;
и незакрашенным квадратом (□) обозначена зависимость от времени концентрации в плазме GPC3 ERY9-1 и GPC3 ERY10-1 соответственно;
на фиг.10 - график, на котором представлена зависимость от времени концентраций в плазме GPC3 ERY9-1 и GPC3 ERY10-1, определенных с использованием экспрессирующих CD3 клеток Ba/F3. Закрашенным ромбом  и незакрашенным квадратом
и незакрашенным квадратом  обозначена зависимость от времени концентрации в плазме GPC3 ERY9-1 и GPC3 ERY10-1 соответственно;
обозначена зависимость от времени концентрации в плазме GPC3 ERY9-1 и GPC3 ERY10-1 соответственно;
на фиг.11 -график, демонстрирующий способность GPC3 BiTE, GPC3 ERY9-1, GPC3 ERY10-1, GPC3 ERY15-1 и катумаксомаба индуцировать цитокины независимым от ракового антигена образом;
на фиг.12 график, демонстрирующий цитотоксичность in vitro GPC3 ERY18 LI, GPC3 ERY18L2, GPC3 ERY18L3, GPC3 ERY18L4 и GPC3 ERY18S1. Закрашенным треугольником 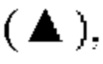 закрашенным кружком
закрашенным кружком 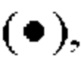 закрашенным квадратом
закрашенным квадратом 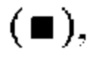 незакрашенным квадратом
незакрашенным квадратом 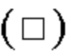 и незакрашенным ромбом
и незакрашенным ромбом  обозначена цитотоксическая активность GPC3 ERY18 LI, GPC3 ERY18 L2, GPC3 ERY18 L3, GPC3 ERY18 L4 и GPC3 ERY18 S1 соответственно;
обозначена цитотоксическая активность GPC3 ERY18 LI, GPC3 ERY18 L2, GPC3 ERY18 L3, GPC3 ERY18 L4 и GPC3 ERY18 S1 соответственно;
на фиг.13 - график, на котором представлено сравнение цитотоксической активности in vitro GPC3 ERY18 L3 и GPC3 ERY10-1. Закрашенным квадратом 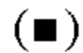 и незакрашенным квадратом (□) обозначена цитотоксическая активность GPC3 ERY18 L3 и GPC3 ERY10-1 соответственно;
и незакрашенным квадратом (□) обозначена цитотоксическая активность GPC3 ERY18 L3 и GPC3 ERY10-1 соответственно;
на фиг.14 график, на котором представлено сравнение цитотоксической активности in vitro GPC3 ERY19-3 и GPC3 BiTE. Незакрашенным квадратом 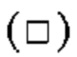 и закрашенным квадратом
и закрашенным квадратом  обозначена цитотоксическая активность GPC3 ERY19-3 и GPC3 BiTE соответственно;
обозначена цитотоксическая активность GPC3 ERY19-3 и GPC3 BiTE соответственно;
на фиг.15А - хроматограмма, демонстрирующая результаты анализа с использованием гель-фильтрации СМ, в которых происходит экспрессия NTAlL/NTAlR/GC33-k0. На фиг.15Б - хроматограмма, демонстрирующая результаты анализа с использованием гель-фильтрации СМ, в которых происходит экспрессия NTA2L/NTA2R/GC33-k0;
на фиг.16 диаграммы, на которых показаны домены, образующие следующие полипептидные комплексы, описанные в примерах, которые представлены в настоящем описании: GPC3 BiTE, GPC3 ERY2, GPC3 ERY5, GPC3 ERY6, GPC3 ERY7, GPC3 ERY8-2, GPC3 ERY9-1, GPC3 ERY 10-1, GPC3 ERY15, GPC3 ERY18 и GPC3 ERY19-3. Домен, заштрихованный перекрестными линиями, обозначает вариабельную область Н-цепи антитела к раковому антигену (GPC3, ЕрСАМ, EGFR); домен с диагональными линиями обозначает вариабельную область L-цепи антитела к раковому антигену (GPC3, ЕрСАМ, EGFR); домен с пунктирными линиями обозначает вариабельную область Н-цепи антитела к CD3; закрашенный домен обозначает вариабельную область L-цепи антитела к CD3; незакрашенный домен обозначает константную область антитела; крестик обозначает молчащую мутацию в Fc и звездочка обозначает мутацию, усиливающую гетеромерную ассоциацию Fc;
на фиг.17 - диаграммы GPC3 BiTE (A); GPC3 ERY 10 (Б); GPC3 ERY2 (В); GPC3 ERY5 (Г); GPC3 ERY6 (Д); GPC3 ERY7 (Е); GPC3 ERY8-2 (Ж); GPC3 ERY9-1 (3); GPC3 ERY10-1 (И); GPC3 ERY15 (К); GPC3 ERY18 (Л) и GPC3 ERY19-3 (М);
на фиг.18 аминокислотные остатки, образующие Fc-домены IgG1, IgG2, IgG3 и IgG4 и их EU-нумерация по Кэботу (в контексте настоящего описания обозначен также как «EU-индекс»);
на фиг.19 - диаграммы, на которых показаны домены, образующие полипептидные комплексы, описанные в примерах, которые представлены в настоящем описании: GPC3 ERY17-2, GPC3 ERY17-3, ЕрСАМ ERY17-2 и ЕрСАМ ERY17-3. Домен, заштрихованный перекрестными линиями, обозначает вариабельную область Н-цепи антитела к раковому антигену (GPC3, ЕрСАМ, EGFR); домен с диагональными линиями обозначает вариабельную область L-цепи антитела к раковому антигену (GPC3, ЕрСАМ, EGFR); домен с пунктирными линиями обозначает вариабельную область Н-цепи антитела к CD3; закрашенный домен обозначает вариабельную область L-цепи антитела к CD3; незакрашенный домен обозначает константную область антитела; крестик обозначает молчащую мутацию в Fc и звездочка обозначает мутацию, усиливающую гетеромерную ассоциацию Fc;
на фиг.20 график, на котором представлено сравнение цитотоксической активности GPC3 BiTE, GPC3 ERY17-2, GPC3 ERY17-3 и GPC3 ERY10-1. Закрашенным квадратом 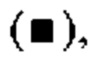 закрашенным треугольником
закрашенным треугольником 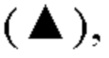 незакрашенным кружком (○) и незакрашенным квадратом (□) обозначена цитотоксическая активность GPC3 BiTE, GPC3 ERY17-2, GPC3 ERY17-3 и GPC3 ERY10-1 соответственно;
незакрашенным кружком (○) и незакрашенным квадратом (□) обозначена цитотоксическая активность GPC3 BiTE, GPC3 ERY17-2, GPC3 ERY17-3 и GPC3 ERY10-1 соответственно;
на фиг.21 - график, демонстрирующий противоопухолевую активность in vivo GPC3 ERY17-2 при моделировании на РС-10 Т-клеточного переноса. Незакрашенным квадратом (□) и закрашенным ромбом 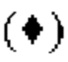 обозначены изменения объема опухолей в группе, для обработки которой применяли GPC3 ERY17-2, и контрольной (обработка ЗФР) группе соответственно;
обозначены изменения объема опухолей в группе, для обработки которой применяли GPC3 ERY17-2, и контрольной (обработка ЗФР) группе соответственно;
на фиг.22 - график, на котором представлено сравнение цитотоксической активности GPC3 ERY17-2 и GPC3 ERY17-2-M20. Закрашенным треугольником 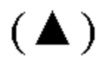 и незакрашенным кружком
и незакрашенным кружком обозначена цитотоксическая активность GPC3 ERY17-2 и GPC3 ERY17-2-M20 соответственно;
обозначена цитотоксическая активность GPC3 ERY17-2 и GPC3 ERY17-2-M20 соответственно;
на фиг.23 график, на котором представлено сравнение цитотоксической активности ЕрСАМ ERY17-2 и ЕрСАМ ERY17-3. Закрашенным треугольником 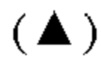 и незакрашенным квадратом
и незакрашенным квадратом 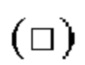 обозначена цитотоксическая активность ЕрСАМ ERY17-2 и ЕрСАМ ERY17-3 соответственно;
обозначена цитотоксическая активность ЕрСАМ ERY17-2 и ЕрСАМ ERY17-3 соответственно;
на фиг.24 диаграммы, на которых показаны домены, образующие полипептидные комплексы, описанные в примерах, которые представлены в настоящем описании: GM1, GM2 и GM0. На «А» показан полипептидный комплекс, в котором регулируется ассоциация на поверхности раздела CH1/CL и интродуцированы модификации типа «выступ-впадина» (KiH). На «Б» показан полипептидный комплекс, в котором не регулируется ассоциация на поверхности раздела CH1/CL или не интродуцированы KiH-модификации. Домен, заштрихованный перекрестными линиями, обозначает вариабельную область Н-цепи антитела к раковому антигену (GPC3, ЕрСАМ, EGFR); домен с диагональными линиями обозначает вариабельную область L-цепи антитела к раковому антигену (GPC3, ЕрСАМ, EGFR); домен с пунктирными линиями обозначает вариабельную область Н-цепи антитела к CD3; закрашенный домен обозначает вариабельную область L-цепи антитела к CD3; незакрашенный домен обозначает константную область антитела; крестик обозначает молчащую мутацию в Fc и звездочка обозначает мутацию, усиливающую гетеромерную ассоциацию Fc;
и имеющий форму пончика символ обозначает мутацию, регулирующую взаимодействие на поверхности раздела CH1/CL;
на фиг.25 - график, на котором представлено сравнение цитотоксической активности GM1, GM2 и GM0. Закрашенным треугольником 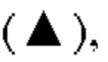 незакрашенным квадратом (□) и незакрашенным кружком
незакрашенным квадратом (□) и незакрашенным кружком  обозначена цитотоксическая активность GM1, GM2 и GM3 соответственно;
обозначена цитотоксическая активность GM1, GM2 и GM3 соответственно;
на фиг.26 график, демонстрирующий цитотоксическую активность EGFR ERY17-2. Закрашенным треугольником  обозначена цитотоксическая активность EGFR ERY17-2.
обозначена цитотоксическая активность EGFR ERY17-2.
Варианты осуществления изобретения
Представленные ниже определения даны с целью объяснения настоящего изобретения. Антитело
В контексте настоящего описания «антитело» относится к встречающемуся в естественных условиях иммуноглобулину или иммуноглобулину, полученному полностью или частично путем синтеза. Антитела можно выделять из встречающихся в естественных условиях источников, таких как встречающиеся в естественных условиях плазма и сыворотка, или из супернатантов культур продуцирующих антитела гибридом. Альтернативно этому антитела можно частично или полностью синтезировать с использованием таких методик, как генетическая рекомбинация. Предпочтительными антителами являются, например, антитела, принадлежащие к какому-либо изотипу иммуноглобулинов или его подклассу. Известные человеческие иммуноглобулины включают антитела следующих девяти классов (изотипов): IgG1, IgG2, IgG3, IgG4, IgAl, IgA2, IgD, IgE и IgM. Из этих изотипов к антителам, предлагаемым в изобретении, относятся IgG1, IgG2, IgG3 и IgG4.
Методы получения антитела с требуемой активностью связывания известны специалистам в данной области. Ниже представлен пример, в котором описан метод получения антитела (антитела к GPC3), связывающегося с GPC3, который принадлежит к семейству GPI-заякоренных рецепторов (Int J Cancer. 103(4), 2003, сс.455-465). Согласно описанному ниже примеру можно получать также антитела, которые связываются с антигеном, отличным от GPC3.
Антитела к GPC3 можно получать в виде поликлональных или моноклональных антител с помощью известных методов. Антитела к GPC3 предпочтительно получали в виде моноклональных антител, выведенных из организма млекопитающих. Указанные выведенные из организма млекопитающих антитела включают антитела, полученные с помощью гибридом или клеток-хозяев, трансформированных экспрессионным вектором, который несет ген антитела, созданным с помощью методов генетической инженерии.
Гибридомы, продуцирующие моноклональные антитела можно получать с использованием известных методов, например, описанных ниже. В частности, млекопитающих иммунизируют с помощью общепринятых методов иммунизации, используя белок GPC3 в качестве сенсибилизирующего антигена. Образовавшиеся иммунные клетки сливают с известными родительскими клетками с помощью общепринятых методов слияния. Затем гибридомы, продуцирующие антитело к GPC3, можно отбирать путем скрининга в отношении продуцирующих моноклональные антитела клеток с помощью общепринятых методов скрининга.
В частности, моноклональные антитела получают согласно описанному ниже методу. Сначала ген GPC3, нуклеотидная последовательность которого представлена в RefSeq под регистрационным номером NM_001164617.1 (SEQ ID NO: 1), можно экспрессировать с получением белка GPC3, последовательность которого представлена в RefSeq под регистрационным номером NP_001158089.1 (SEQ ID NO: 2), который можно применять в качестве сенсибилизирующего антигена для получения антитела. Для этого генную последовательность, кодирующую GPC3, встраивают в известный экспрессионный вектор и соответствующие клетки-хозяева трансформируют этим вектором. Требуемый человеческий белок GPC3 очищают из клеток-хозяев или супернатантов культур с помощью известных методов. Например, для получения растворимого GPC3 из супернатантов культур удаляют путем делеции аминокислоты в положениях 564-580, которые формируют гидрофобную область, соответствующую GPI-заякоривающей последовательности, применяемой для заякоривания GPC3 на клеточной мембране, из полипептидной последовательности GPC3 SEQ ID NO: 2, а затем образовавшийся белок экспрессируют вместо белка GPC3, имеющего SEQ ID NO: 2. В альтернативном варианте в качестве сенсибилизирующего антигена можно применять очищенный встречающийся в естественных условиях белок GPC3.
Очищенный белок GPC3 можно применять в качестве сенсибилизирующего антигена для иммунизации млекопитающих. В качестве сенсибилизирующего антигена можно применять также неполный пептид GPC3. В этом случае неполный пептид можно получать химическим синтезом на основе аминокислотной последовательности человеческого GPC3 или путем встраивания части гена GPC3 в экспрессионный вектор для экспрессии. В альтернативном варианте неполный пептид можно получать путем расщепления белка GPC3 протеазой. Конкретные варианты осуществления изобретения не накладывают ограничения на длину и область неполного пептида GPC3. Предпочтительную область можно произвольно выбирать из числа аминокислотных остатков аминокислотной последовательности, находящихся в положениях 564-580 в аминокислотной последовательности SEQ ID NO: 2. Количество аминокислот, образующих пептид, который можно применять в качестве сенсибилизирующего агента, предпочтительно составляет по меньшей мере пять или более, шесть или более или семь или более. Более конкретно, в качестве сенсибилизирующего антигена можно применять пептид, состоящий из 8-50 остатков, более предпочтительно из 10-30 остатков.
В альтернативном варианте в качестве сенсибилизирующего антигена можно применять слитый белок, полученный путем слияния требуемого неполного полипептида или пептида белка GPC3, с другим полипептидом. Например, для получения слитых белков, предназначенных для применения в качестве сенсибилизирующих антигенов, предпочтительно применяют Fc-фрагменты антитела и пептидные метки. Векторы для экспрессии указанных слитых белков можно конструировать путем слияния в рамке считывания генов, кодирующих два или большее количество требуемых полипептидных фрагментов, и встраивания слитого гена в экспрессионный вектор, описанный выше. Методы получения слитых белков описаны в Molecular Cloning, 2-ое изд. (Sambrook J. и др., Molecular Cloning, 2-ое изд. 1989, 9.47-9.58, изд-во Cold Spring Harbor Lab. Press). Методы получения GPC3, предназначенного для применения в качестве сенсибилизирующего антигена, и методы иммунизации с использованием GPC3 конкретно описаны в WO 2003/000883, WO 2004/022754 и WO 2006/006693.
Отсутствует конкретное ограничение, касающееся млекопитающих, подлежащих иммунизации с помощью сенсибилизирующего антигена. Однако предпочтительно выбирать млекопитающих с учетом их совместимости с родительскими клетками, применяемыми для клеточного слияния. В целом, предпочтительно применяют грызунов, таких как мыши, крысы, а также хомяки, кролики, и обезьян.
Вышеуказанных животных иммунизируют сенсибилизирующим антигеном с помощью известных методов. Общепринятыми методами иммунизации являются, например, внутрибрюшинная или подкожная инъекция млекопитающих сенсибилизирующим антигеном. В частности, сенсибилизирующий антиген можно соответствующим образом разводить в ЗФР (забуференный фосфатом физиологический раствор), физиологическом соляном растворе или т.п. При необходимости с антигеном смешивают общепринятый адъювант, такой как полный адъювант Фрейнда, и смесь эмульгируют.Затем сенсибилизирующий антиген вводят млекопитающему несколько раз с 4-21-дневными интервалами. При иммунизации сенсибилизирующим антигеном можно использовать соответствующие носители. В частности, когда в качестве сенсибилизирующего антигена используют низкомолекулярный неполный пептид, то иногда для иммунизации требуется сочетать пептид, представляющий собой сенсибилизирующий антиген, с белком-носителем, таким как альбумин или гемоцианин лимфы улитки.
Альтернативно этому можно получать продуцирующие требуемое антитело гибридомы с помощью описанной ниже ДНК-иммунизации. ДНК-иммунизация представляет собой метод иммунизации, который обеспечивает иммуностимуляцию посредством экспрессии сенсибилизирующего антигена в организме иммунизированного животного в результате введения ДНК-вектора, сконструированного таким образом, чтобы он обеспечивал экспрессию гена, кодирующего антигенный белок, в организме животного. По сравнению с общепринятыми методами иммунизации, при которых животным, подлежащим иммунизации, вводят белковый антиген, ДНК-иммунизация, по-видимому, имеет следующие преимущества:
- можно осуществлять иммуностимуляцию, сохраняя при этом структуру мембранного белка, такого как GPC3; и
- отсутствует необходимость в очистке антигена для иммунизации. Для получения моноклонального антитела, предлагаемого в настоящем
изобретении, с использованием ДНК-иммунизации сначала вводят животному, подлежащему иммунизации, ДНК, экспрессирующую белок GPC3. ДНК, кодирующую GPC3, можно синтезировать с помощью известных методов, таких как ПЦР. Полученную ДНК встраивают в соответствующий экспрессионный вектор и затем его вводят животному, подлежащему иммунизации. Предпочтительно применяемые для этой цели экспрессионные векторы включают, например, поступающие в продажу экспрессионные векторы, такие как pcDNA3.1. Векторы можно вводить в организм с помощью общепринятых методов. Например, ДНК-иммунизацию осуществляют с использованием генной пушки для интродукции золотых частиц, покрытых экспрессионным вектором, в клетки тела животного, подлежащего иммунизации. Антитела, распознающие GPC3, можно получать также методами, описанными в WO 2003/104453.
После описанной выше иммунизации млекопитающего у него подтверждают в сыворотке повышенный титр GPC3-связывающего антитела. После этого получают из организма млекопитающего иммунные клетки и затем используют их для клеточного слияния. В частности, в качестве иммунных клеток предпочтительно применяют спленоциты.
Клетку миеломы млекопитающих применяют в качестве клетки, подлежащей слиянию с вышеуказанным иммуноцитом. Клетки миеломы предпочтительно содержат приемлемый маркер селекции для скрининга. Маркер селекции придает клеткам характеристики, обеспечивающие их выживание (или гибель) в специфических условиях культивирования. В качестве маркера селекции известны дефицит гипоксантин-гуанин-фосфорибозилтрансферазы (сокращенно обозначенный далее в контексте настоящего описания как дефицит HGPRT) и дефицит тимидинкиназы (сокращенно обозначенный далее в контексте настоящего описания как дефицит TK). Клетки с дефицитом HGPRT или ТК обладают чувствительностью к гипоксантин-аминоптерин-тимидину (сокращенно обозначена далее в контексте настоящего описания как ГАТ-чувствительность). Клетки с ГАТ-чувствительностью не могут синтезировать ДНК в селекционной ГАТ-среде и в результате погибают.Однако, когда клетки сливают со здоровыми клетками, они могут продолжать синтез ДНК с использованием «реутилизационного» пути здоровых клеток, и в результате они могут расти даже в селекционной ГАТ-среде.
Клетки с HGPRT-дефицитом и TK-дефицитом можно отбирать в среде, содержащей 6-тиогуанин, 8-азагуанин (сокращенно обозначенный далее в контексте настоящего описания как 8AG) или 5'-бромдезоксиуридин соответственно. Здоровые клетки уничтожаются, поскольку они включают эти пиримидиновые аналоги в их ДНК. При этом клетки с дефицитом этих ферментов могут выживать в селекционной среде, поскольку они не могут включать указанные пиримидиновые аналоги. Кроме того, к маркеру селекции относится устойчивость к G418, которая обеспечивается геном устойчивости к неомицину, придающим устойчивость к 2-дезоксистрептаминовым антибиотикам (аналоги гентамицина). Известны различные типы клеток миелом, которые можно применять для клеточного слияния.
Например, в качестве клеток миеломы предпочтительно можно применять следующие клетки:
P3(P3x63Ag8.653) (J. Immunol. 123 (4), 1979, сс. 1548-1550);
P3x63Ag8U.1 (Current Topics in Microbiology and Immunology 81, 1978, cc. 1-7);
NS-1 (C. Eur. J. Immunol. 6 (7), 1976, cc. 511-519);
MPC-11 (Cell 8 (3), 1976, cc. 405-415);
SP2/0 (Nature 276 (5685), 1978, cc. 269-270);
FO (J. Immunol. Methods 35 (1-2), 1980, cc. 1-21);
S194/5.XX0.BU.1 (J. Exp.Med. 148 (1), 1978, cc. 313-323);
R210 (Nature 277 (5692), 1979, cc. 131-133) и т.д.
Клеточное слияние иммуноцитов и клеток миеломы, как правило, осуществляют с помощью известных методов, например, метода, описанного у Kohler и Milstein и др. (Methods Enzymol. 73, 1981, cc. 3-46).
Более конкретно, клеточное слияние можно осуществлять, например, в общепринятой культуральной среде в присутствии усиливающего клеточное слияние агента. Усиливающие клеточное слияние агенты включают, например, полиэтиленгликоль (ПЭГ) и вирус Сендай (гемагглютинирующий японский вирус мышей) (HVJ). При необходимости для повышения эффективности слияния добавляют также вспомогательную субстанцию, такую как диметилсульфоксид.
Соотношение иммуноцитов и клеток миеломы можно определять по усмотрению исследователя, предпочтительно, например, одна клетка миеломы на каждые 1-10 иммуноцитов. Культуральные среды, применяемые для клеточных слияний, включают, например, среды, пригодные для выращивания клеточных линий миелом, такие как среда RPMI1640 и среда MEM, а также другая общепринятая культуральная среда, применяемая для данного типа клеточной культуры. Кроме того, в культуральную среду предпочтительно можно добавлять добавки в виде сыворотки, такой как фетальная телячья сыворотка (FCS).
Для клеточного слияния указанные выше иммунных клети и клетки миеломы, взятые в предварительно определенных количествах, хорошо перемешивают в указанной выше культуральной среде. Затем к ней добавляют предварительно нагретый до температуры примерно 37°С раствор ПЭГ (например, средняя молекулярная масса которого составляет примерно от 1000 до 6000) в концентрации, составляющей, как правило, от 30 до 60% (мас./об.). Смесь осторожно перемешивают до получения требуемых слитых клеток (гибридомы). Затем указанную выше культуральную среду постепенно добавляют к клеткам и повторно центрифугируют для удаления супернатанта. Таким путем можно удалять агенты для клеточного слияния, которые являются нежелательными для роста гибридом.
Полученные таким образом гибридомы можно отбирать путем культивирования, используя общепринятую селективную среду, например, ГАТ-среду (культуральная среда, содержащая гипоксантин, аминоптерин и тимидин). Клетки, отличные от требуемых гибридом (неслитые клетки), можно уничтожать путем последующего культивирования в вышеуказанной ГАТ-среде в течение достаточного периода времени. Как правило, период составляет от нескольких дней до нескольких недель. Затем осуществляют скрининг гибридом, продуцирующих требуемое антитело, и по отдельности клонируют с помощью общепринятых методов серийных разведений.
Полученные таким образом гибридромы можно отбирать, используя селекционную среду на основе маркера селекции, который несут клетки миеломы, применяемые для клеточного слияния. Например, клетки с HGPRT-или ТК-дефицитом можно отбирать путем культивирования с использованием ГАТ-среды (культуральная среда, содержащая гипоксантин, аминоптерин и тимидин). А частности, когда для клеточного слияния используют чувствительные к ГАТ клетки миеломы, то клетки, для которых характерно успешное слияние со здоровыми клетками, могут избирательно размножаться в ГАТ-среде. Клетки, отличные от требуемых гибридом (неслитые клетки) можно уничтожать путем культивирования в вышеуказанной ГАТ-среде в течение достаточного периода времени. В частности, требуемые гибридомы можно отбирать путем культивирования, как правило, в течение периода времени, составляющего от нескольких дней до нескольких недель. Затем осуществляют скрининг гибридом, продуцирующих требуемое антитело, и по отдельности клонируют с помощью общепринятых методов серийных разведений.
Требуемые антитела предпочтительно можно отбирать и по отдельности клонировать с помощью методов скрининга, основанных на известной реакции антиген/антитело. Например, GPC3-связывающее моноклональное антитело может связываться с GPC3, экспрессируемым на клеточной поверхности. Можно осуществлять скрининг указанных моноклональных антител с помощью метода разделения клеток на основе возбуждения флуоресценции (FACS). FACS представляет собой систему, которая позволяет оценивать связывание антитела с клеточной поверхностью посредством анализа клеток, контактирующих с флуоресцентным антителом, с использованием лазерного пучка, и путем оценки флуоресценции, испускаемой индивидуальными клетками.
Для скрининга с использованием FACS в отношении гибридом, которые продуцируют моноклональное антитело, предлагаемое в настоящем изобретении, прежде всего получают экспрессирующие GPC3 клетки. Клетки, которые предпочтительно используют для скрининга, представляют собой клетки млекопитающих, в которых происходит принудительная экспрессия GPC3. В качестве контроля можно избирательно определять с использованием нетрансформированных клеток млекопитающих в качестве клеток-хозяев способность антитела связываться с расположенным на клеточной поверхности GPC3. В частности, гибридомы, продуцирующие моноклональное антитело к GPC3, можно выделять путем отбора гибридом, которые продуцируют антитело, связывающееся с клетками, принудительно экспрессирующими GPC3, но не с клетками-хозяевами.
Альтернативно этому способность антитела связываться с иммобилизованными экспрессирующими GPC3 клетками можно оценивать на основе принципа ELISA. Например, экспрессирующие GPC3 клетки иммобилизуют на лунках планшета для ELISA. Супернатанты культур гибридом приводят в контакт с иммобилизованными клетками в лунках и выявляют антитела, которые связываются с иммобилизованными клетками. Когда моноклональные антитела имеют мышиное происхождение, то антитела, связанные с клетками, можно выявлять с помощью антитела к мышиному иммуноглобулину. Гибридомы, продуцирующие требуемое антитело, которое обладает антигенсвязывающей активностью, отбирают путем описанного выше скрининга, и их можно клонировать методом серийных разведений или сходным методом.
Полученные таким путем гибридомы, продуцирующие моноклональные антитела, можно пересевать в общепринятую культуральную среду и хранить в жидком азоте в течение длительного периода времени.
Указанные выше гибридомы культивируют с помощью общепринятого метода и требуемые моноклональные антитела можно получать из супернатантов культур. Альтернативно этому гибридомы интродуцируют и выращивают в пригодных для этой цели млекопитающих и моноклональные антитела получают из асцитов. Первый метод пригоден для получения антител с высокой степенью чистоты.
Предпочтительно можно применять также антитела, кодируемые генами антител, которые клонированы из продуцирующих антитела клеток, таких как указанные выше гибридомы. Клонированный ген антитела встраивают в соответствующий вектор и его интродуцируют в хозяина для экспрессии кодируемого геном антитела. Методы выделения генов антител, встраивания генов в векторы и трансформации клеток-хозяев разработаны ранее (см., например, Vandamme и др., Eur. J. Biochem. 192(3), 1990, сс. 767-775). Методы получения рекомбинантных антител также известны и описаны ниже.
Например, кДНК, кодирующую вариабельную область (V-область) антитела к GPC3, получают из клеток гибридомы, экспрессирующих антитело к GPC3. Для этой цели сначала из гибридом экстрагируют общую РНК. Методы, которые применяют для экстракции мРНК из клеток, включают, например:
- метод ультрацентрифугирования в присутствии гуанидина (Biochemistry 18(24), 1979, сс. 5294-5299) и
- AGPC-метод (Anal. Biochem. 162(1), 1987, сс. 156-159).
Экстрагированные мРНК можно очищать с помощью набора для очистки мРНК (фирма GE Healthcare Bioscience) или аналогичного набора. В качестве альтернативы можно применять также поступающие в продажу наборы для экстракции мРНК непосредственно из клеток, такие как набор для очистки мРНК QuickPrep (фирма GE Healthcare Bioscience). мРНК можно получать из гибридом с использованием таких наборов. Кодирующие V-область антитела кДНК можно синтезировать из полученных мРНК с помощью обратной транскриптазы. кДНК можно синтезировать с помощью набора для синтеза первой цепи кДНК, содержащего обратную транскриптазу AMV (Reverse Transcriptase First-strand cDNA Synthesis Kit) (фирма Seikagaku Co.) или аналогичного набора. Кроме того, для синтеза и амплификации кДНК можно применять набор для амплификации кДНК SMART RACE (фирма Clontech) и основанный на ПЦР 5'-RACE-(быстрая амплификация концов кДНК)-метод (Proc. Natl. Acad. Sci. USA 85(23), 1988, cc. 8998-9002; Nucleic Acids Res. 17(8), 1989, cc. 2919-2932). При таком процессе синтеза кДНК соответствующие описанные ниже сайты, распознаваемые рестриктазами, можно интродуцировать на оба конца кДНК.
Представляющий интерес фрагмент кДНК очищают из полученного ПНР-продукта и затем его встраивают путем лигирования в ДНК-вектор. Таким путем создают рекомбинантный вектор и интродуцируют в Е. coli или подобного хозяина. После селекции колоний требуемый рекомбинантный вектор можно получать из колониеобразующих Е. coli. Затем с использованием известного метода, такого как метод терминации нуклеотидной цепи, определяют, имеет ли рекомбинантный вектор представляющую интерес нуклеотидную последовательность кДНК.
5'-RACE-метод, в котором используют праймеры для амплификации гена вариабельной области, как правило, применяют для выделения гена, кодирующего вариабельную область. Сначала конструируют библиотеку кДНК, применяемую для 5'-RACE (5'-RACE-библиотека кДНК) с использованием РНК, экстрагированных из клеток гибридомы, в качестве матрицы. Для синтеза 5'-RACE-библиотеки кДНК можно использовать поступающий в продажу набор, такой как набор для амплификации кДНК SMART RACE.
Ген антитела амплифицируют с помощью ПЦР, используя полученную 5'-RACE-библиотеку кДНК в качестве матрицы. Праймеры для амплификации гена мышиного антитела можно создавать на основе известных генных последовательностей антител. Нуклеотидные последовательности праймеров варьируются в зависимости от подкласса иммуноглобулина. Таким образом, предпочтительно предварительно определять подкласс с помощью доступного набора, такого как набор для изотипирования мышиных моноклональных антител Iso Strip (Iso Strip mouse monoclonal antibody isotyping kit) (фирма Roche Diagnostics).
В частности, например, для выделения генов, кодирующих мышиный IgG, применяют праймеры, которые обеспечивают амплификацию генов, кодирующих тяжелые цепи γl, γ2а, γ2b и γ3 и легкие цепи κ и λ. В целом, праймер, сайт гибридизации («отжига») которого с константной областью расположен вблизи вариабельной области, применяют в качестве 3'-концевого праймера для амплификации гена вариабельной области IgG. При этом праймер, присоединенный к набору конструкций 5' RACE-библиотеки кДНК, применяют в качестве 5'-концевого праймера.
Амплифицированные таким образом ПЦР-продукты применяют для реконструирования иммуноглобулинов, состоящих из комбинации тяжелых и легких цепей. Требуемое антитело можно отбирать, используя в качестве показателя GPC3-связывающую активность реконструированного иммуноглобулина. Например, когда задачей является выделение антитела к GPC3, то более предпочтительно, чтобы связывание антитела с GPC3 являлось специфическим. Можно осуществлять скрининг GPC3-связывающих антител, например, с использованием следующих стадий, на которых:
(1) приводят в контакт экспрессирующую GPC3 клетку с антителом, содержащим V-область, которая кодируется кДНК, выделенной из гибридомы;
(2) определяют связывания антитела с экспрессирующей GPC3 клеткой; и
(3) отбирают антитело, которое связывается с экспрессирующей GPC3 клеткой.
Методы определения связывания антитела с экспрессирующими GPC3 клетками, являются известными. В частности, связывание антитела с экспрессирующими GPC3 клетками можно определять с помощью описанных выше методик, таких как FACS. Иммобилизованные образцы экспрессирующих GPC3 клеток можно применять для оценки связывающей активности антитела.
Предпочтительные методы скрининга антител, в которых используют связывающую активность в качестве показателя, включают также методы пэннинга, основанные на использовании фаговых векторов. Методы скрининга с использованием фаговых векторов имеют преимущество, когда гены антитела выделяют из библиотек подкласса тяжелой цепи и легкой цепи из популяции клеток, экспрессирующих поликлональные антитела. Гены, кодирующие вариабельные области тяжелой цепи и легкой цепи, можно сшивать с помощью приемлемой линкерной последовательности с образованием одноцепочечного Fv (scFv). Фаги, презентующие на своей поверхности scFv, можно получать путем встраивания гена, кодирующего scFv, в фаговый вектор. Фаги приводят в контакт с представляющим интерес антигеном. Затем ДНК, кодирующую scFv, который обладает представляющей интерес связывающей активностью, можно выделять путем сбора фагов, связанных с антигеном. Указанный процесс можно повторять при необходимости для обогащения scFv, которые обладают представляющей интерес связывающей активностью.
После выделения кДНК, кодирующей V-область представляющего интерес антитела к GPC3, кДНК расщепляют рестриктазами, которые распознают сайты рестрикции, интродуцированные в оба конца кДНК. Предпочтительные рестриктазы распознают и расщепляют нуклеотидную последовательность, которая встречается с низкой частотой в нуклеотидной последовательности гена антитела. Кроме того, для встраивания однокопийного расщепленного фрагмента в правильной ориентации предпочтительно интродуцируют в представляющий интерес вектор сайт рестрикции для фермента, который образует «липкий» конец. Кодирующую V-область антитела к GPC3 кДНК расщепляют согласно описанному выше методу и встраивают в приемлемый экспрессионный вектор для создания экспрессионного вектора антитела. В том случае, когда ген, кодирующий константную область (С-область) антитела, и ген, кодирующий указанную выше V-область, сливают в рамке считывания, то получают химерное антитело. В контексте настоящего описания «химерное антитело» означает, что константная область и вариабельная область отличаются по своему происхождению. Так, помимо мышиных/человеческих гетерохимерных антител к химерным антителам, предлагаемым в настоящем изобретении, относятся также человеческие/человеческие аллохимерные антитела. Экспрессионный вектор химерного антитела можно создавать путем встраивания указанного выше гена V-области в экспрессионный вектор, который уже содержит константную область. В частности, например,
последовательность, распознаваемую рестриктазой, которая вырезает указанный выше ген V-области, предпочтительно следует помещать в 5'-область экспрессионного вектора, несущего ДНК, которая кодирует константную область (С-область) требуемого антитела. Экспрессионный вектор химерного антитела создают путем слияния в рамке считывания двух генов, расщепленных одной и той же комбинацией рестриктаз.
Для получения моноклонального антитела к GPC3 гены антитела встраивают в экспрессионный вектор таким образом, чтобы экспрессия генов происходила под контролем регулирующей экспрессию области. Регулирующая экспрессию область, предназначенная для экспрессии антител, включают, например, энхансеры и промоторы. Кроме того, соответствующую сигнальную последовательность можно присоединять к аминоконцу таким образом, чтобы антитело секретировалось из клеток наружу. В описанных ниже примерах в качестве сигнальной последовательности применяют пептид, который имеет аминокислотную последовательность MGWSCIILFLVATATGVHS (SEQ ID NO: 72). Наряду с ней можно присоединять другие приемлемые сигнальные последовательности. Экспрессированный полипептид расщепляется на карбоксильном конце указанной выше последовательности и образовавшийся полипептид секретируется из клеток наружу в виде зрелого полипептида. Затем приемлемые клетки-хозяева трансформируют экспрессионным вектором и получают рекомбинантные клетки, экспрессирующие ДНК, которая кодирует антитело к GPC3.
ДНК, которые кодируют тяжелую цепь антитела (Н-цепь) и легкую цепь антитела (L-цепь), встраивают по отдельности в различные экспрессионные векторы для экспрессии гена антитела. Молекулу антитела, имеющую Н- и L-цепи, можно экспрессировать, осуществляя для этой цели контрансфекцию одной и той же клетки-хозяина векторами, в которые встроены соответственно гены Н-цепи и L-цепи. В качестве альтернативы, клетки-хозяева можно трансформировать одним экспрессионным вектором, в который встроены ДНК, кодирующие Н- и L-цепи (см. WO 94/11523).
Известны различные комбинации клеток-хозяев/экспрессионных векторов для получения антитела путем интродукции выделенных генов антител в соответствующих хозяев. Все эти системы экспрессии можно применять для выделения антигенсвязывающих доменов и CD3-связанных доменов, предлагаемых в настоящем изобретении. Приемлемыми эукариотическими клетками, которые применяют в качестве клеток-хозяев, являются клетки животных, клетки растений и клетки грибов. В частности, клетки животных представляют собой, например, следующие клетки:
(1) клетки млекопитающих: СНО, COS, миеломы, почки детеныша хомяка (BHK), HeLa, Vero или т.п.;
(2) клетки амфибий: ооциты шпорцевой лягушки (Xenopus) или т.п. и
(3) клетки насекомых: sf9, sf21, Tn5 или т.п.
Кроме того, в качестве растительной клетки известна система экспрессии генов антитела, в которой используют клетки, полученные из представителей рода Nicohana, например, Nicotiana tabacum. Для трансформации растительных клеток можно применять культивированные клетки каллуса.
Кроме того, следующие клетки можно применять в качестве грибных клеток:
дрожжи: рода Saccharomyces, например, Saccharomyces cerevisiae, и рода Pichia, например, Pichia pastoris, и
нитчатые грибы: рода Aspergillus, например, Aspergillus niger.
Кроме того, известны системы экспрессии генов антитела, в которых используют прокариотические клетки. Например, согласно настоящему изобретению можно применять бактериальные клетки, т.е. клетки Е. coli, клетки Bacillus subtilis и т.п. Экспрессионные векторы, которые несут представляющие интерес гены антитела, интродуцируют в эти клетки путем трансфекции. Трансфектированные клетки культивируют in vitro, и требуемое антитело можно получать из культуры трансформированных клеток.
Помимо указанных выше клеток-хозяев для получения рекомбинантного антитела можно применять также трансгенных животных. Это означает, что антитело можно получать из животного, в организм которого интродуцирован ген, кодирующий представляющее интерес антитело. Например, ген антитела можно создавать в виде слитого гена посредством встраивания в рамке считывания в ген, кодирующего белок, который специфически образуется в молоке. В качестве белка, секретируемого в молоко, можно применять, например, козий β-казеин. ДНК-фрагменты, содержащие слитый ген, в который входит ген антитела, инъецируют в эмбрион козы и затем этот эмбрион интродуцируют в самку козы. Требуемые антитела можно получать в виде белка, слитого с молочным белком из молока трансгенной козы, родившейся от козы, являющейся реципиентом эмбриона (или ее потомства). Кроме того, для увеличения объема молока, содержащего требуемое антитело, которое продуцируется трансгенной козой, трансгенной козе можно при необходимости вводить гормоны (Ebert K. М. и др., Bio/Technology 12 (7), 1994, сс. 699-702).
Когда полипептидный комплекс, представленный в настоящем описании, вводят человеку, то в качестве антигенсвязывающего домена комплекса можно применять также антигенсвязывающий домен, выведенный из антитела, полученного путем генетической рекомбинации, которое искусственно модифицировано для снижения гетерологичной антигенности в отношении человека и других животных. Указанные антитела, полученные путем генетической рекомбинации, включают, например, гуманизированные антитела. Эти модифицированные антитела можно получать с помощью известных методов.
Вариабельная область антитела, применяемая для получения антигенсвязывающего домена полипептидного комплекса, представленного в настоящем описании, как правило, состоит из трех гипервариабельных участков (CDR), разделенных четырьмя каркасными участками (FR). CDR представляет собой область, которая в значительной степени определяет специфичность связывания антитела. Для аминокислотных последовательностей CDR характерна высокая степень вариабельности. С другой стороны, образующие FR аминокислотные последовательности часто обладают высокой идентичностью даже среди антител с различными специфичностями связывания. Таким образом, как правило, путем трансплантации CDR специфичность связывания конкретного антитела можно интродуцировать в другое антитело.
Гуманизированное антитело называют также реконструированным человеческим антителом. В частности, известны гуманизированные антитела, полученные путем трансплантации CDR антитела животного кроме человека, такого как мышиное антитело, в человеческое антитело и т.п. Известны также общепринятые методики генной инженерии, предназначенные для получения гуманизированных антител. В частности, например, ПЦР с перекрывающимися праймерами представляет собой известный метод трансплантации CDR мышиного антитела в человеческий FR. При осуществлении ПЦР с перекрывающимися праймерами нуклеотидную последовательность, которая кодирует CDR мышиного антитела, подлежащий трансплантации, добавляют к праймерам, предназначенным для синтеза FR человеческого антитела. Получают праймеры для каждого из четырех FR. Принято считать, что когда осуществляют трансплантацию мышиного CDR в человеческий FR, то для поддержания функции CDR целесообразно выбирать человеческий FR, обладающий высоким уровнем идентичности с мышиным FR. Таким образом, как правило, является предпочтительным применять человеческий FR, который содержит аминокислотную последовательность, обладающую высоким уровнем идентичности с аминокислотной последовательностью FR, который примыкает к подлежащему трансплантации мышиному CDR.
Нуклеотидные последовательности, подлежащие лигированию, создают таким образом, чтобы они были соединены друг с другом в рамке считывания. Человеческие FR синтезируют индивидуально с использованием соответствующих праймеров. В результате получают продукты, в которых ДНК, кодирующая мышиный CDR, присоединена к индивидуальным ДНК, кодирующим FR. Нуклеотидные последовательности, кодирующие мышиный CDR каждого продукта, создают таким образом, чтобы они перекрывались друг с другом. Затем осуществляют реакцию синтеза комплементарной цепи для «отжига» перекрывающихся CDR-участков продуктов, синтезированных с использованием гена человеческого антитела в качестве матрицы. С помощью этой реакции человеческие FR встраивают путем лигирования через мышиные CDR-последовательности.
Полноразмерный ген V-области, в которую, в конце концов, лигированы три CDR и четыре FR, амплифицируют с использованием праймеров, гибридизующихся с 5'- или 3'-концом, которые добавляют с последовательностями, распознаваемыми приемлемыми рестриктазами. Экспрессионный вектор для гуманизированного антитела можно получать путем встраивания полученной согласно описанному выше методу ДНК и ДНК, которая кодирует С-область человеческого антитела, в экспрессионный вектор таким образом, чтобы лигировать их в рамке считывания. После трансфекции хозяина рекомбинантным вектором для создания рекомбинантных клеток рекомбинантные клетки культивируют и ДНК, кодирующую гуманизированное антитело, экспрессируют с получением гуманизированного антитела в культуре клеток (см. публикацию европейского патента ЕР 239400 и публикацию международной заявки на патент WO 1996/002576).
Путем качественной или количественной оценки и измерения антигенсвязывающей активности гуманизированного антитела, полученного согласно описанному выше методу, можно отбирать FR человеческого антитела, которые позволяют CDR образовывать предпочтительный антигенсвязывающий центр при лигировании посредством CDR. Аминокислотные остатки в FR при необходимости можно заменять так, чтобы CDR реконструированного человеческого антитела образовывали приемлемый антигенсвязывающий центр. Например, мутации аминокислотной последовательности можно интродуцировать в FR, используя ПЦР-метод, применяемый для трансплантации мышиного CDR в человеческий FR. Более конкретно, мутации неполной нуклеотидной последовательности можно интродуцировать в праймеры, гибридизующиеся с FR. Мутации нуклеотидной последовательности интродуцируют в FR, синтезированные с использованием указанных праймеров. Мутантные последовательности FR, имеющие требуемые характеристики, можно отбирать путем измерения и оценки активности мутантного антитела с аминокислотной заменой в отношении связывания с антигеном с помощью упомянутого выше метода (Sato K. и др., Cancer Res. 53, 1993, сс.85 1-856).
В качестве альтернативы, требуемые человеческие антитела можно получать путем иммунизации трансгенных животных, имеющих полный спектр генов человеческого антитела (см. WO 1993/012227; WO 1992/003918; WO 1994/002602; WO 1994/025585; WO 1996/034096; WO 1996/033735), с использованием ДНК-иммунизации.
Кроме того, известны методики получения человеческих антител путем пэннинга с использованием библиотек человеческих антител. Например, V-область человеческого антитела экспрессируют в виде одноцепочечного антитела (scFv) на поверхности фага с использованием метода фагового дисплея. Можно отбирать фаги, экспрессирующие scFv, который связывается с антигеном. Последовательность ДНК, кодирующую V-область человеческого антитела, которая связывается с антигеном, можно определять путем анализа генов отобранных фагов. Определяют последовательность ДНК scFv, который связывается с антигеном. Экспрессионный вектор получают путем слияния последовательности V-области в рамке считывания с последовательностью С-области требуемого человеческого антитела и последующего встраивания в приемлемый экспрессионный вектор. Экспрессионный вектор интродуцируют в клетку, пригодную для указанной выше экспрессии. Человеческое антитело можно получать путем экспрессии гена, кодирующего человеческое антитело, в клетках. Такие методы уже описаны (см. WO 1992/001047; WO 1992/020791; WO 1993/006213; WO 1993/011236; WO 1993/019172; WO 1995/001438; WO 1995/015388).
Антигенсвязывающий домен
В контексте настоящего описания «антигенсвязывающий домен» относится к участку антитела, содержащему область, которая специфически связывается и является комплементарной полному антигену или его участку. Когда молекулярная масса антигена является большой, то антитело может связываться только с конкретным участком антигена. Указанный конкретный участок называют «эпитопом». Антигенсвязывающий домен может быть образован одним или несколькими вариабельными доменами антитела. Предпочтительно антигенсвязывающие домены содержат как вариабельную область легкой цепи (VL) антитела, так и вариабельную область тяжелой цепи (VH) антитела. Указанные предпочтительные антигенсвязывающие домены включают, например, «одноцепочечный Fv (scFv)», «одноцепочечное антитело», «Fv», «одноцепочечный Fv2 (scFv2)», «Fab» и «F(ab')2».
Антигенсвязывающие домены полипептидных комплексов, предлагаемых в настоящем изобретении, могут связываться с одним и тем же эпитопом. Эпитоп может присутствовать в белке, который содержит аминокислотную последовательность SEQ ID NO: 2 или 4. Альтернативно этому, антигенсвязывающие домены полипептидных комплексов, предлагаемых в настоящем изобретении, могут индивидуально связываться с различными эпитопами. Эпитоп может присутствовать в белке, который содержит аминокислотную последовательность SEQ ID NO: 2 или 4. Специфичность
«Специфичность» означает, что молекула, которая связывается специфически с одним или несколькими партнерами по связыванию, не обладает какой-либо значимой способностью связываться с молекулами, отличными от указанных партнеров. Кроме того, понятие «специфичность» используют также, когда антигенсвязывающий домен является специфическим в отношении конкретного эпитопа из нескольких эпитопов, входящих в антиген. Когда эпитоп, с которым связывается антигенсвязывающий домен, входит в несколько различных антигенов, то полипептидный комплекс, содержащий антигенсвязывающий домен, может связываться с различными антигенами, которые содержат эпитоп.
Антиген
Настоящее описание не ограничено каким-либо конкретным антигеном, и можно применять любой антиген за исключением CD3. Указанные антигены включают, например, рецепторы, раковые антигены, антигены ГКГ и дифференцированные антигены. Рецепторы включают рецепторы, принадлежащие к семейству рецепторов гематопоэтического фактора роста, семейству цитокиновых рецепторов, семейству тирозинкиназных рецепторов, семейству серин/треонинкиназных рецепторов, семейству TNF-рецепторов, семейству рецепторов, связанных с G-белками, семейству GPI-заякоренных рецепторов, семейству тирозинфосфатазных рецепторов, семейству факторов адгезии и семейству рецепторов гормонов. Рецепторы, принадлежащие к этим семействам рецепторов, и их характеристики описаны в различных документах, например, в обзорах, таких как New Comprehensive Biochemistry под ред. Cooke В. A., King R.J.В., van der Molen H.J., т.18B «Hormones and their Actions Part II», 1988, cc. 1-46, изд-во) Elsevier Science Publishers BV.; и SAIBO KOGAKU (Cell Technology) Supplementary Volume Handbook Series «Secchaku Inshi Handbook (Handbook for Adhesion factors)» под ред. M. Miyasaka, 1994, Shujunsha, Tokyo, Japan; и у Patthy, Cell, 61(1), 1990, сс.13-14; Ullrich и др., Cell 61(2), 1990, сс.203-212; Massague, Cell 69(6), 1992, сс.1067-1070; Miyajima и др., Annu. Rev. Immunol. 10, 1992, сс.295-331; Taga и др., FASEB J. 6, 1992, сс.3387-3396); Fantl и др., Annu. Rev. Biochem. 62, 1993, cc. 453-481; Smith и др., Cell 76(6), 1994, сс.959-962, Flower D.R. в Biochim. Biophys. Acta: Flower Biochim. Biophys. Acta, 1422(3), 1999, cc. 207-234.
В частности, рецепторы, принадлежащие к указанным выше семействам рецепторов, предпочтительно включают, например, рецепторы человеческого и мышиного эритропоэтина (ЕРО) (Blood 76(1), 1990, сс.31-35; Cell 57(2), 1989, сс.277-285), рецепторы человеческого и мышиного гранулоцитарного колониестимулирующего фактора (G-CSF) (Proc. Natl. Acad. Sci. USA. 87(22), 1990, cc. 8702-8706; mG-CSFR, Cell 61(2), 1990, cc. 341-350), рецепторы человеческого и мышиного тромбопоэтина (ТРО) (Proc. Natl. Acad. Sci. USA 89(12), 1992, cc. 5640-5644; EMBO J. 12(7), 1993, cc. 2645-53), рецепторы человеческого и мышиного инсулина (Nature 313(6005), 1985, сс.756-761), рецепторы человеческого и мышиного лиганда Flt-3 (Proc. Natl. Acad. Sci. USA 91(2), 1994, cc. 459-463), рецепторы человеческого и мышиного тромбоцитарного фактора роста (PDGF) (Proc. Natl. Acad. Sci. USA. 85(10), 1988, cc. 3435-3439), рецепторы человеческого и мышиного интерферона (IFN)-α и -β (Cell 60(2), 1990, сс.225-234; и Cell 77(3), 1994, сс.391-400), рецепторы человеческого и мышиного лептина, рецепторы человеческого и мышиного гормона роста (GH), рецепторы человеческого и мышиного интерлейкина (IL)-10, рецепторы человеческого и мышиного инсулиноподобного фактора роста (IGF)-I, рецепторы человеческого и мышиного лейкозингибирующего фактора (LIF) и рецепторы человеческого и мышиного цилиарного нейротрофического фактора (CNTF).
Раковые антигены представляют собой антигены, которые экспрессируются в том случае, когда клетки становятся злокачественными, и их обозначают также как «специфические для опухоли антигены». Кроме того, аномальные сахарные цепи, которые экспрессируются на клеточной поверхности или молекулах белков, когда клетки становятся раковыми, также представляют собой раковые антигены и их обозначают как «ассоциированные с раком углеводные антигены». Указанные раковые антигены включают, например, GPC3 (Int J Cancer. 103(4), 2003, cc. 455-465), Ер С AM (Proc. Natl. Acad. Sci. USA. 86(1), 1989, cc. 27-31), EGFR, CA19-9, CA15-3 и сиалил88ЕА-1 (SLX). GPC3 принадлежит к указанному выше семейству GPI-заякоренных рецепторов, и он экспрессируется при нескольких типах рака, включая рак печени. ЕрСАМ экспрессируется при множестве видов рака, включая рак легкого, и его полинуклеотидные и полипептидные последовательности представлены в базе данных RefSeq под регистрационным номерами NM_002354.2 (SEQ ID NO: 3) и NP_002345.2 (SEQ ID NO: 4) соответственно.
Как правило, антигены ГКГ подразделяют на антигены ГКГ класса I и класса П. Антигены ГКГ класса I включают HLA-A, -В, -С, -Е, -F, -G и -Н, а антигены ГКГ класса II включают HLA-DR, -DQ и -DP.
Дифференцированные антигены включают CD1, CD2, CD4, CD5, CD6, CD7, CD8, CD10, CD11a, CD11b, CD11c, CD13, CD14, CD15s, CD16, CD18, CD19, CD20, CD21, CD23, CD25, CD28, CD29, CD30, CD32, CD33, CD34, CD35, CD38, CD40, CD41a, CD41b, CD42a, CD42b, CD43, CD44, CD45, CD45RO, CD48, CD49a, CD49b, CD49c, CD49d, CD49e, CD49f, CD51, CD54, CD55, CD56, CD57, CD58, CD61, CD62E, CD62L, CD62P, CD64, CD69, CD71, CD73, CD95, CD102, CD106, CD122, CD126 и CDw130.
Эпитоп
«Эпитоп» означает антигенную детерминанту в антигене и относится к антигенному сайту, с которым связывается антигенсвязывающий домен полипептидного комплекса, представленного в настоящем описании. Так, например, эпитоп можно характеризовать на основе его структуры. Альтернативному этому, эпитоп можно характеризовать на основе антигенсвязывающей активности полипептидного комплекса, который распознает эпитоп.Когда антиген представляет собой пептид или полипептид, то эпитоп можно определять по аминокислотным остаткам, образующим эпитоп.Альтернативно этому, когда эпитоп представляет собой сахарную цепь, то эпитоп можно определять по специфической для него структуре сахарной цепи.
Линейный эпитоп представляет собой эпитоп, который содержит эпитоп, у которого распознается первичная аминокислотная последовательность. Указанный линейный эпитоп, как правило, содержит по меньшей мере три и наиболее часто по меньшей мере пять, например, примерно от 8 до 10 или от 6 до 20 аминокислотных остатков в определенной последовательности.
В отличие от линейного эпитопа «конформационный эпитоп» представляет собой эпитоп, в котором первичная аминокислотная последовательность, составляющая эпитоп, не является единственной детерминантой распознаваемого эпитопа (например, не является обязательным, чтобы первичная аминокислотная последовательность конформационного эпитопа распознавалась специфическим в отношении эпитопа антителом). Конформационные эпитопы могут содержать большее количество аминокислотных остатков по сравнению с линейными эпитопами. Распознающее конформационный эпитоп антитело распознает трехмерную структуру пептида или белка. Например, когда белковая молекула уложена и образует трехмерную структуру, аминокислоты и/или полипептидные основные цепи, которые образуют конформационный эпитоп, выравниваются, и эпитоп становится распознаваемым для антитела. Методы определения конформаций эпитопов включают (но, не ограничиваясь только ими), например, рентгеновскую кристаллографию, двухмерный ядерный магнитный резонанс, сайтспецифическое спиновое мечение и электронный парамагнитный резонанс (см., например, Epitope Mapping Protocols in Methods in Molecular Biology, под ред. Morris, т. 66, 1996).
Примеры методов оценки связывания эпитопа тестируемым полипептидным комплексом, содержащим домен, связывающий антиген GPC3, описаны ниже. Можно применять также описанные ниже в качестве примеров методы оценки связывания эпитопа тестируемым полипептидным комплексом, содержащим антигенсвязывающий домен для антигена, отличного от GPC3.
Например, для подтверждения того, что тестируемый полипептидный комплекс, содержащий домен, связывающий антиген GPC3, распознает линейный эпитоп в молекуле GPC3, можно применять описанный ниже метод. Для указанной выше цели синтезируют линейный пептид, содержащий аминокислотную последовательность, образующую внеклеточный домен GPC3. Пептид можно синтезировать химически или получать с помощью методов генной инженерии, используя область в кДНК GPC3, которая кодирует аминокислотную последовательность, соответствующую внеклеточному домену.
Затем тестируемый полипептидный комплекс, содержащий домен, связывающий антиген GPC3, оценивают в отношении его активности связывания с линейным пептидом, который содержит аминокислотную последовательность, образующую внеклеточный домен. Например, иммобилизованный линейный пептид можно применять в качестве антигена при осуществлении ELISA для оценки активности в отношении связывания полипептидного комплекса с пептидом. Альтернативно этому, активность связывания с линейным пептидом можно оценивать по уровню ингибирования линейным пептидом связывания полипептидного комплекса с экспрессирующими GPC3 клетками. Эти анализы могут демонстрировать активность связывания полипептидного комплекса с линейным пептидом.
Обладает ли полипептидный комплекс, содержащий домен, связывающий антиген GPC3, способностью распознавать конформационный эпитоп, можно определять следующим образом. Для указанной выше цели получают экспрессирующие GPC3 клетки. Считается, что тестируемый полипептидный комплекс, содержащий домен, связывающий антиген GPC3, распознает конформационный эпитоп, если он при контакте отличается сильным связыванием с экспрессирующими GPC3 клетками, но незначительно связывается с иммобилизованным линейным пептидом, который содержит аминокислотную последовательность, образующую внеклеточный домен GPC3. В контексте настоящего описания «незначительно связывается» означает, что активность связывания составляет 80% или менее, как правило, 50% или менее, предпочтительно 30% или менее и наиболее предпочтительно 15% или менее по сравнению с активностью связывания с клетками, экспрессирующими человеческий GPC3.
Методы оценки активности связывания тестируемого полипептидного комплекса, содержащего домен, связывающий антиген GPC3, с экспрессирующими GPC3 клетками включают, например, методы, описанные в: Antibodies: A Laboratory Manual (под ред. Harlow, David Lane, изд-во Cold Spring Harbor Laboratory, 1988, cc. 359-420). В частности, оценку можно осуществлять на основе принципа ELISA или метода разделения клеток на основе возбуждения флуоресценции (FACS) с использованием в качестве антигена экспрессирующих GPC3 клеток.
В формате ELISA активность связывания тестируемого полипептидного комплекса, содержащего домен, связывающий антиген GPC3, с экспрессирующими GPC3 клетками можно оценивать количественно путем сравнения уровней сигналов, возникающих в процессе ферментативной реакции. В частности, тестируемый полипептидный комплекс добавляют в планшет для ELISA, на котором иммобилизованы экспрессирующие GPC3 клетки. Затем тестируемый полипептидный комплекс, связанный с клетками, выявляют с помощью меченного ферментом антитела, которое распознает тестируемый полипептидный комплекс.Альтернативно этому, когда применяют FACS, приготавливают серийные разведения тестируемого полипептидного комплекса и титр антитела, связывающегося с экспрессирующими GPC3 клетками, можно определять путем сравнения активности связывания с экспрссирующими GPC3 клетками.
Связывание тестируемого полипептидного комплекса с антигеном, который экспрессируется на поверхности клеток, суспендированных в буфере или в сходной среде, можно выявлять с помощью проточного цитометра. Известными проточными цитометрами являются, например, следующие устройства:
FACSCanto™ II,
FACSAria™,
FACSArray™,
FACSVantage™ SE
FACSCalibur™ (все товарные знаки фирмы BD Biosciences),
EPICS ALTRA HyPerSort,
Cytomics FC 500,
EPICS XL-MCL ADC EPICS XL ADC,
Cell Lab Quanta/Cell Lab Quanta SC (все товарные знаки фирмы Beckman Coulter).
Предпочтительными методами оценки активности связывания тестируемого полипептидного комплекса, содержащего домен, связывающий антиген GPC3, с антигеном является, например, следующий метод. Сначала экспрессирующие GPC3 клетки подвергают взаимодействию с тестируемым полипептидным комплексом, а затем его окрашивают меченным с помощью ФИТЦ вторичным антителом, которое распознает полипептидный комплекс.Тестируемый полипептидный комплекс соответствующим образом разводят приемлемым буфером с получением комплекса в требуемой концентрации. Например, комплекс можно применять в концентрации, составляющей от 10 мкг/мл до 10 нг/мл. Затем определяют интенсивность флуоресценции и количество клеток с помощью FACSCalibur (фирма BD). Интенсивность флуоресценции, определенная с помощью анализов на основе программы CELL QUEST (фирма BD), а именно, выраженная в виде средних геометрических значений, отражает уровень связывания антитела с клетками. Это означает, что активность связывания тестируемого полипептидного комплекса, которая характеризуется количеством связанного тестируемого полипептидного комплекса, можно оценивать, определяя среднее геометрическое значение.
Имеет ли тестируемый полипептидный комплекс, содержащий домен, связывающий антиген GPC3, общий эпитоп с другим полипептидным комплексом, можно оценивать по конкуренции между двумя комплексами в отношении одного и того же эпитопа. Конкуренцию между полипептидными комплексами можно определять путем анализа перекрестной блокады или с помощью сходного анализа. Например, предпочтительным анализом перекрестной блокады является конкурентный ELISA-анализ.
В частности, при осуществлении анализа перекрестной блокады белок GPC3, иммобилизованный в лунках титрационного микропланшета, предварительно инкубируют в присутствии полипептидного комплекса, рассматриваемого в качестве конкурента-кандидата, или без него, а затем вносят тестируемый полипептидный комплекс.Количество связанного с белком GPC3 тестируемого полипептидного комплекса в лунках находится в обратной корреляции с активностью связывания полипептидного комплекса, рассматриваемого в качестве конкурента-кандидата, который конкурирует за связывание с одним и тем же эпитопом. Это означает, что чем выше аффинность полипептидного комплекса, рассматриваемого в качестве конкурента-кандидата, к одному и тому же эпитопу, тем ниже активность связывания тестируемого полипептидного комплекса с сенсибилизированными белком GPC3 лунками.
Количество тестируемого полипептидного комплекса, связанного через белок GPC3 с лунками, легко определять путем предварительного мечения полипептидного комплекса. Например, меченный биотином полипептидный комплекс оценивают с использованием конъюгата авидин/пероксидаза и соответствующего субстрата. В частности, анализ перекрестной блокады, в котором применяют в качестве меток ферменты, такие как пероксидаза, называют «ELISA-анализом в конкурентных условиях (конкурентный анализ)». Полипептидный комплекс можно метить также с помощью других предназначенных для мечения субстанций, которые можно выявлять или оценивать. В частности, известны радиоактивные метки, флуоресцентные метки и т.п.
Когда полипептидный комплекс, рассматриваемый в качестве конкурента-кандидата, может блокировать связывание тестируемого полипептидного комплекса, который содержит домен, связывающий антиген GPC3, по меньшей мере на 20%, предпочтительно по меньшей мере на 20-50% и предпочтительно по меньшей мере на 50% по сравнению с активностью связывания, установленной в контрольном эксперименте, который проводят в отсутствии полипептидного комплекса, рассматриваемого в качестве конкурента-кандидата, то считается, что для тестируемого полипептидного комплекса характерна выраженная способность к связыванию с тем же эпитопом, с которым связывается полипептидный комплекс, рассматриваемый в качестве конкурента-кандидата, или он конкурирует за связывание с тем же самым эпитопом.
Если структура эпитопа, связанного с тестируемым полипептидным комплексом, который содержит домен, связывающий антиген GPC3, уже идентифицирована, то для решения вопроса о том, имеют ли тестируемый и контрольный полипептидные комплексы один и тот же эпитоп, можно сравнивать активности связывания двух полипептидных комплексов с пептидом, полученным путем интродукции аминокислотных мутаций в пептид, образующий эпитоп.
Для оценки указанных выше активностей связывания, например, сравнивают активности связывания тестируемого и контрольного полипептидных комплексов с линейным пептидом, в который интродуцирована мутация, с помощью указанного выше формата ELISA. Помимо метода ELISA активность связывания в отношении мутантного пептида, связанного с колонкой, можно определять, пропуская через колонку тестируемый и контрольный полипептидные комплексы и затем оценивая количество полипептидного комплекса, элюированного в раствор для элюции. Известны методы адсорбции мутантного пептида на колонке, например, в форме слитого с GST пептида.
Альтернативно этому, когда идентифицированный эпитоп представляет собой конформационный эпитоп, то для решения вопроса о том, имеют ли тестируемый и контрольный полипептидные комплексы общий эпитоп, можно применять следующий метод. Сначала получают экспрессирующие GPC3 клетки и клетки, экспрессирующие GPC3 с мутацией, интродуцированной в эпитоп.Тестируемый и контрольный полипептидные комплексы добавляют в клеточную суспензию, полученную путем суспендирования этих клеток в приемлемом буфере, таком как ЗФР. Затем клеточные суспензии соответствующим образом отмывают буфером и добавляют меченное с помощью ФИТЦ антитело, которое распознает тестируемый и контрольный полипептидные комплексы. Определяют интенсивность флуоресценции и количество клеток, окрашенных меченым антителом, используя FACSCalibur (фирма BD). Тестируемый и контрольный полипептидные комплексы соответствующим образом разводят приемлемым буфером и применяют в требуемой концентрации. Например, их можно применять в концентрации от 10 мкг/мл до 10 нг/мл. Интенсивность флуоресценции определяют с помощью анализа, для оценки результатов которого применяют программу CELL QUEST (фирма BD), а именно, определяют среднее геометрическое значение, которое отражает количество меченого антитела, связанного с клетками. Это означает, что активности связывания тестируемого и контрольного полипептидных комплексов, которые характеризуются количеством связанного меченого антитела, можно определять, оценивая среднее геометрическое значение.
При осуществлении описанного выше метода для решения вопроса о том, характерно ли для полипептидного комплекса «незначительное связывание с клетками, которые экспрессируют мутантный GPC3», можно применять, например, следующий метод. Сначала тестируемый и контрольный полипептидные комплексы, связанные с клетками, которые экспрессируют мутантный GPC3, окрашивают меченым антителом. Затем определяют интенсивность флуоресценции в клетках. Когда для оценки флуоресценции используют проточный питометр, то интенсивность флуоресценции можно анализировать с помощью программы CELL QUEST. На основе средних геометрических значений в присутствии полипептидного комплекса и без него можно рассчитывать согласно приведенной ниже формуле используемое для сравнения значение (AGeo-Mean), характеризующее степень увеличения интенсивности флуоресценции в результате связывания с полипептидным комплексом.
ΔGeo-Mean = Geo-Mean (в присутствии полипептидного комплекса)/Geo-Mean (в отсутствии полипептидного комплекса)
Используемое для сравнения среднее геометрическое значение (значение AGeo-Mean для мутантной молекулы GPC3), определенное с помощью описанного выше анализа, которое отражает количество тестируемого полипептидного комплекса, связанного с клетками, которые экспрессируют мутантный GPC3, сравнивают с относительным значением ΔGeo-Mean, которое отражает количество тестируемого полипептидного комплекса, связанного с экспрессирующими GPC3 клетками. В этом случае концентрации тестируемого полипептидного комплекса, применяемые для определения относительных значений ΔGeo-Mean для экспрессирующих GPC3 клеток и клеток, экспрессирующих мутантный GPC3, наиболее предпочтительно регулируют таким образом, чтобы они были одинаковыми или практически одинаковыми. Полипептидный комплекс, для которого подтверждена способность распознавать эпитоп в GPC3, применяют в качестве контрольного полипептидного комплекса.
Если используемое для сравнения значение ΔGeo-Mean тестируемого полипептидного комплекса в отношении клеток, экспрессирующих мутантный GPC3, ниже чем используемое для сравнения значение ΔGeo-Mean тестируемого полипептидного комплекса в отношении клеток, экспрессирующих GPC3, по меньшей мере на 80%, предпочтительно на 50%, более предпочтительно на 30% и наиболее предпочтительно на 15%, то считают, что тестируемый полипептидный комплекс «незначительно связывается с клетками, которые экспрессируют мутантный GPC3». Формула определения значения Geo-Mean (среднее геометрическое значение) описана в руководстве по применению программы CELL QUEST (фирма BD biosciences). Когда путем сравнения установлено, что относительные значения являются практически эквивалентными, то можно считать, что тестируемый и контрольный полипептидные комплексы имеют одинаковый эпитоп.Вариабельный фрагмент (Fv)
В контексте настоящего описания понятие «вариабельный фрагмент (Fv)» относится к минимальной единице выведенного из антитела антигенсвязывающего домена, который состоит из пары, включающей вариабельную область легкой цепи антитела (VL) и вариабельную область тяжелой цепи антитела (VH). В 1988 г. Skerra и Pluckthun обнаружили, что гомогенные и активные антитела можно получать из периплазматической фракции Е. coli путем встраивания гена антитела по ходу транскрипции относительно бактериальной сигнальной последовательности и индукции экспрессии гена в Е. coli (Science 240(4855), 1988, cc. 1038-1041). В Fv, полученном из периплазматической фракции, VH ассоциируется с VL таким образом, чтобы связываться с антигеном.
Согласно настоящему описанию Fv предпочтительно включают, например, пару Fv, которые представляют собой полипептидный комплекс или т.п., содержащий:
(1) двухвалентный антигенсвязывающий домен, который представляет собой двухвалентный scFv, в котором один одновалентный scFv двухвалентного scFv сцеплен с одним полипептидом, образующим Fc-домен, посредством Fv-фрагмента тяжелой цепи, образующего CD3-связывающий домен, а другой одновалентный scFv сцеплен с другим полипептидом, образующим Fc-домен, посредством Fv-фрагмента легкой цепи, образующего CD3-связывающий домен;
(2) домен, содержащий Fc-домен, который не обладает способностью связывать Fcγ-рецептор и который получен из аминокислот, образующих Fc-домен IgG1, IgG2a, IgG3 или IgG4, и
(3) по меньшей мере один одновалентный CD3-связывающий домен, в котором Fv-фрагменты легкой цепи и тяжелой цепи в результате
ассоциации образуют такой CD3-связывающий домен, который может связываться с антигеном CD3.
scFv, одноцепочечное антитело и sc(Fv)2
В контексте настоящего описания понятия «scFv», «одноцепочечное антитело» и «sc(Fv)2» все относятся к фрагменту антитела в виде одной полипептидной цепи, которая содержит вариабельные области, выведенные из тяжелой и легкой цепей, но не содержит константную область. Как правило, одноцепочечное антитело содержит также полипептидный линкер между VH- и VL-доменами, позволяющий образовывать требуемую структуру, которая, вероятно, обеспечивает связывание антигена. Одноцепочечное антитело подробно описано у Pluckthun в: «The Pharmacology of Monoclonal Antibodies», т. 113, под ред. Rosenburg и Moore, изд-во Springer-Verlag, New York, 1994, cc. 269-315» (см. также публикацию международного патента WO 1988/001649; US №№4946778 и 5260203). В конкретном варианте осуществления изобретения одноцепочечное антитело может быть биспецфическим и/или гуманизированным.
scFv представляет собой антигенсвязывающий домен, в котором VH и VL, образующие Fv, сцеплены друг с другом с помощью пептидного линкера (Proc.Natl. Acad. Sci. U.S.A. 85(16), 1988, cc. 5879-5883). VH и VL могут удерживаться в непосредственной близости с помощью пептидного линкера.
sc(Fv)2 представляет собой одноцепочечное антитело, в котором четыре вариабельные области двух VL и двух VH сцеплены линкерами такими как пептидные линкеры, с образованием одной цепи (J Immunol. Methods 231(1-2), 1999, cc. 177-189). Две VH и две VL можно выводить из различных моноклональных антител. Указанные sc(Fv)2 предпочтительно включают, например, биспецифический sc(Fv)2, который распознает два эпитопа, присутствующих в одном антигене, что описано в Journal of Immunology 152(11), 1994, cc. 5368-5374. sc(Fv)2 можно получать методами, известными специалистам в данной области. Например, sc(Fv)2 можно получать путем связывания scFv с помощью линкера, такого как пептидный линкер.
Согласно настоящему описанию к форме антигенсвязывающего домена, образующего sc(Fv)2, относится антитело, в котором две единицы VH и две единицы VL организованы в следующем порядке: VH, VL, VH и VL ([VH]-линкер-[VL]-линкер-[VH]-линкер-[VL]), начиная с N-конца одноцепочечного полипептида. Порядок расположения двух единиц VH и двух единиц VL не ограничен указанной выше формой, и их можно организовывать в любом
порядке. Пример порядка расположения в различных формах приведен ниже.
[VL]-линкер-[VH]-линкер-[VH]-линкер-[VL]
[VH]-линкер-[VL]-линкер-[VL]-линкер-[VH]
[VH]-линкер-[VHJ-линкер-[VL]-линкер-[VL]
[VL]-линкер-[VL]-линкер-[VH]-линкер-[VH]
[VL]-линкер-[VH]-линкер-[VL]-линкер-[VH].
Молекулярная форма sc(Fv)2 подробно описана также в WO 2006/132352. Таким образом, согласно указанным описаниям специалисты в данной области могут получать требуемый sc(Fv)2, предназначенный для создания полипептидных комплексов, представленных в настоящем описании.
Кроме того, полипептидные комплексы, предлагаемые в настоящем изобретении, можно конъюгировать с полимером-носителем, таким как ПЭГ, или органическим соединением, таким как противораковое средство. Альтернативно этому, дополнительную последовательность сахарной цепи предпочтительно встраивают в полипептидные комплексы для того, чтобы сахарная цепь обеспечивала требуемое действие.
Линкеры, предназначенные для связывания вариабельных областей антитела, представляют собой произвольные пептидные линкеры, которые можно интродуцировать с помощью генной инженерии, синтетические линкеры и линкеры, описанные, например, в, Protein Engineering, 9(3), 1996, сс.299-305. Однако для целей настоящего изобретения наиболее предпочтительными являются пептидные линкеры. Длина пептидных линкеров специально не ограничена, и специалисты в данной области могут выбирать ее в зависимости от поставленной задачи. Предпочтительная длина составляет пять или большее количество аминокислот (при этом, верхний предел составляет (но, не ограничиваясь только указанным), как правило, вплоть до 30 аминокислот или менее, предпочтительно 20 аминокислот или менее), и наиболее предпочтительно 15 аминокислот.Когда sc(Fv)2 содержит три пептидных линкера, то их длина может быть одинаковой или различной.
Например, указанные пептидные линкеры включают:
Ser,
Gly⋅Ser,
Gly⋅Gly⋅Ser,
Ser⋅Gly⋅Gly,
Gly⋅Gly⋅Gly⋅Ser (SEQ ID NO: 5),
Ser⋅Gly⋅Gly⋅Gly (SEQ ID NO: 6),
Gly⋅Gly⋅Gly⋅Gly⋅Ser (SEQ ID NO: 7),
Ser⋅Gly⋅Gly⋅Gly⋅Gly (SEQ ID NO: 8),
Gly⋅Gly⋅Gly⋅Gly⋅Gly⋅Ser (SEQ ID NO: 9),
Ser⋅Gly⋅Gly⋅Gly⋅Gly⋅Gly (SEQ ID NO: 10),
Gly⋅Gly⋅Gly⋅Gly⋅Gly⋅Gly⋅Ser (SEQ ID NO: 11),
Ser⋅Gly⋅Gly⋅Gly⋅Gly⋅Gly⋅Gly (SEQ ID NO: 12),
(Gly⋅Gly⋅Gly⋅Gly⋅Ser (SEQ ID NO: 7))n,
(Ser⋅Gly⋅Gly⋅Gly⋅Gly (SEQ ID NO: 8))n,
где n обозначает целое число от 1 или выше. Специалисты в данной области могут выбирать длину или последовательности пептидных линкеров в зависимости от поставленной задачи.
Как правило, для перекрестного сшивания используют синтетические линкеры (химические перекрестносшивающие агенты), они представляют собой, например:
N-гидроксисукцинимид (NHS),
дисукцинимидилсуберат (DSS),
бис(сульфосукцинимидил)суберат (BS3),
дитиобис(сукцинимидилпропионат) (DSP),
дитиобис(сульфосукцинимидилпропионат) (DTSSP),
этиленгликольбис(сукцинимидилсукцинат) (EGS),
этиленгликольбис(сульфосукцинимидилсукцинат) (сульфо-EGS),
дисукцинимидилтартрат (DST), дисульфосукцинимидилтартрат (сульфо-DST),
бис[2-(сукцинимидоксикарбонилокси)этил]сульфон (BSOCOES) и
и бис[2-(сульфосукцинимидоксикарбонилокси)этил]сульфон (сульфо-BSOCOES). Эти перекрестносшивающие агенты поступают в продажу.
Как правило, требуется три линкера для соединения вместе четырех вариабельных областей антитела. Применяемые линкеры могут быть одного типа или различных типов.
Fab, F(ab')2 и Fab'
«Fab» состоит из одной легкой цепи и СН1-домена и вариабельной области из одной тяжелой цепи. Тяжелая цепь молекулы Fab не может образовывать дисульфидные мостки с тяжелой цепью другой молекулы.
«F(ab')2» или «Fab» получают обработкой иммуноглобулина (моноклональное антитело) протеазой, такой как пепсин и папаин, и они относятся к фрагменту антитела, получаемого расщеплением иммуноглобулина (моноклональное антитело) вблизи дисульфидных мостиков, присутствующих между шарнирными областями в каждой из двух Н-цепей. Например, папаин расщепляет IgG перед дисульфидными мостиками, присутствующими между шарнирными областями в каждой из двух Н-цепей, с образованием двух гомологичных фрагментов антитела, в которых L-цепь, содержащая VL (вариабельная область L-цепи), и CL (константная область L-цепи), сцеплены с фрагментом Н-цепи, содержащим VH (вариабельный домен Н-цепи) и CHγ1 (γl-область в константной области Н-цепи), через дисульфидный мостик в их С-концевых областях. Каждый из указанных двух гомологичных фрагментов антител обозначают как Fab'.
«F(ab')2» состоит из двух легких цепей и двух тяжелых цепей, содержащих в константной области СН1-домен и часть СН2-доменов, в результате между двумя тяжелыми цепями образуются дисульфидные мостики. F(ab')2, образующий полипептидный комплекс, представленный в настоящем описании, предпочтительно можно получать следующим образом. Полное моноклональное антитело или сходное антитело, содержащее требуемый антигенсвязывающий домен, частично расщепляют протеазой, такой как пепсин; и Fc-фрагменты удаляют путем адсорбции на колонке с белком А. Не существует ограничения касательно конкретной протеазы, если она обладает способностью избирательно расщеплять полное антитело с образованием F(ab')2 в соответствующих для данного фермента реакционных условиях, таких как значение рН. Указанные протеазы представляют собой, например, пепсин и фицин.
Fc-домен
Fc-домен, который образует полипептидный комплекс, представленный в настоящем описании, предпочтительно можно получать следующим образом. Антитело, такое как моноклональное антитело, частично расщепляют протеазой, такой как пепсин. Затем образовавшийся фрагмент адсорбируют на колонке с белком А или белком G и элюируют соответственным буфером для элюции. Не существует ограничения касательно конкретной протеазы, если она обладает способностью избирательно расщеплять антитела, такие как моноклональные антитела, в соответствующих для данного фермента реакционных условиях, таких как значение рН. Указанные протеазы представляют собой, например, пепсин и фицин.
Полипептидные комплексы, представленные в настоящем описании, содержат Fc-домен с пониженной способностью связывать Fcγ- рецептор, которые включают аминокислоты, образующие Fc-домен IgG1, IgG2, IgG3 или IgG4.
Изотип антитела определяют на основе структуры константной области. Константные области изотипов IgG1, IgG2, IgG3 и IgG4 обозначают как Cγ1, Сγ2, Сγ3 и Сγ4 соответственно. Аминокислотные последовательности Fc-домена полипептидов, которые образуют человеческие Cγ1, Сγ2, Сγ3 и Сγ4, в качестве примера представлены в SEQ ID NO: 23, 24, 25 и 26 соответственно. Взаимосвязь между аминокислотными остатками, образующими каждую аминокислотную последовательность и EU-нумерацией Кэбота (обозначена в контексте настоящего описания как EU-индекс), представлена на фиг.18.
Понятие «Fc-домен» относится к области вне F(ab')2, которая содержит две легкие цепи и две тяжелые цепи, содержащие часть константной области, которая включает СН1-домен и область между СН1- и СН2-доменами, что позволяет образовываться дисульфидным мостикам между двумя тяжелыми цепями. Fc-домен, образующий полипептидный комплекс, представленный в настоящем описании, предпочтительно можно получать следующим образом. Моноклональное антитело в виде IgG1, IgG2, IgG3 или IgG4 или сходное антитело частично расщепляют протеазой, такой как пепсин, с последующей элюцией фракции, адсорбированной на колонке с белком А. Не существует ограничения касательно конкретной протеазы, если она обладает способностью избирательно расщеплять полное антитело с образованием F(ab')2 в соответствующих для данного фермента реакционных условиях, таких как значение рН. Указанные протеазы представляют собой, например, пепсин и фицин.
Fcγ-рецептор
Понятие «Fcγ-рецептор» относится к рецептору, обладающему способностью связываться с Fc-доменом моноклональных антител в виде IgG1, IgG2, IgG3 или IgG4, и оно включает всех представителей, принадлежащих к семейству белков, которые кодируются главным образом геном Fcγ-рецептора. У человека семейство включает FcγRI (CD64), включая изоформы FcγRIa, FcγRIb и FcγRIc; FcγRII (CD32), включая изоформы FcγRIIa (в том числе аллотип Н131 и R131), FcγRIIb (включая FcγRIIb-1 и FcγRIIb-2) и FcγRIIc; и FcγRIII (CD16), включая изоформу FcγRIIIa (в том числе аллотипы V158 и F158), и FcγRIIIb (в той числе аллотипы FcγRIIIb-NA1 и FcγRIIIb-NA2); а также все неидентифицированные человеческие FcγR, изоформы FcγR и их аллотипы. Однако Fcγ-рецептор не ограничен указанными примерами. FcγR включает (но не ограничиваясь только ими) FcγR, выведенные из антител человека, мышей, крыс, кроликов и обезьян. FcγR можно выводить из любого организма. Мышиный FcγR включает (но, не ограничиваясь только ими) FcγRI (CD64), FcγRII (CD32), FcγRIII (CD16) и FcγRIII-2 (CD16-2), а также все неидентифицированные мышиные FcγR, изоформы FcγR и их аллотипы. Указанные предпочтительные Fcγ-рецепторы включают, например, человеческие FcγI (CD64), FcγIIA (CD32), FcγIIB (CD32), FcγIIIA (CD16) и/или FcγIIIB (CD16). Полинуклеотидная последовательность и аминокислотная последовательность FcγI представлены в SEQ ID NO: 13 (NM_000566.3) и 14 (NP_000557.1) соответственно; полинуклеотидная последовательность и аминокислотная последовательность FcγIIA представлены в SEQ ID NO: 15 (ВС020823.1) и 16 (ААН20823.1) соответственно; полинуклеотидная последовательность и аминокислотная последовательность FcγIIB представлены в SEQ ID NO: 17 (ВС146678.1) и 18 (AAI46679.1) соответственно; полинуклеотидная последовательность и аминокислотная последовательность FcγIIIA представлены в SEQ ID NO: 19 (ВС033678.1) и 20 (ААН33678.1) соответственно и полинуклеотидная последовательность и аминокислотная последовательность FcγIIIB представлены в SEQ ID NO: 21 (ВС128562.1) и 22 (AAI28563.1) соответственно (в скобках указан регистрационный номер каждой последовательности в базе данных RefSeq). Обладает ли Fcγ-рецептор способностью связываться с Fc-доменом моноклонального антитела IgG1, IgG2, IgG3 или IgG4, можно оценивать с помощью ALPHA Screen®-анализа (гомогенный анализ усиленной за счет эффекта близости люминисценции (Amplified Luminescent Proximity Homogeneous Assay)), BIACORE-метода на основе резонанса поверхностного плазмона (SPR) и др. (Proc. Natl. Acad. Sci. USA 103(11), 2006, cc. 4005-4010), помимо описанных выше форматов FACS и ELISA.
При этом понятие «Fc-лиганд» или «эффекторный лиганд» относится к молекуле и предпочтительно полипептиду, который связывается с Fc-доменом антитела, образуя комплекс Fc/Fc-лиганд. Молекулу можно получать из любого организма. Связывание Fc-лиганда с Fc предпочтительно индуцирует одну или несколько эффекторных функций. Указанные лиганды включают (но, не ограничиваясь только ими) Fc-рецепторы, FcγR, FcaR, FcsR, FcRn, C1q и С3, маннансвязывающий лектин, маннозный рецептор, белок A Staphylococcus, белок G Staphylococcus и вирусные FcγR. Fc-лиганды включают также гомологи Fc-рецептора (FcRH) (Davis и др., Immunological Reviews 190, 2002, cc. 123-136), которые представляют собой семейство Fc-рецепторов, гомологичных FcγR. К Fc-лигандам относятся также неидентифицированные молекулы, которые связываются с Fc.
Активность связывания с Fcγ-рецептором
Нарушение активности связывания Fc-домена с любым из Fcγ-рецепторов FcγI, FcγIIA, FcγIIB, FcγIIIA и/или FcγIIIB можно оценивать, используя описанные выше форматы FACS и ELISA, а также ALPHA Screen-анализ (гомогенный анализ усиленной за счет эффекта близости люминисценции, BIACORE-метод на основе резонанса поверхностного плазмона (SPR) (Ргос.Natl. Acad. Sci. USA 103(11), 2006, cc. 4005-4010).
ALPHA Screen-анализ осуществляют на основе технологии ALPHA, которая основана на описанном ниже принципе, с использованием двух типов гранул. Люминисцентный сигнал становится обнаруживаемым только тогда, когда происходит биологическое взаимодействие молекул, связанных с гранулами-донорами, с молекулами, связанными с гранулами-акцепторами, и когда обе гранулы находятся в непосредственной близости друг от друга. Возбужденный лазерным пучком фотосенсибилизатор в гранулах-донорах превращает кислород в возбужденный синглетный кислород. Когда синглетный кислород диффундирует из гранул-доноров и достигает гранул-акцепторов, локализованных в непосредственной близости, то индуцируется хемилюминисцентная реакция в гранулах-акцепторах. В итоге эта реакция приводит к испусканию света. Если молекулы, связанные с гранулами-донорами, не взаимодействуют с гранулами-акцепторами, то синглетный кислород, который продуцируется гранулами-донорами, не достигает гранул-акцепторов и хемилюминисцентная реакция не происходит.
Например, меченный биотином полипептидный комплекс иммобилизуют на гранулах-донорах, а меченный глутатион-8-трансферазой (GST) Fcγ-рецептор иммобилизуют на гранулах-акцепторах. В отсутствии полипептидного комплекса, содержащего конкурирующий мутантный Fc-домен, Fcγ-рецептор взаимодействует с полипептидным комплексом, содержащим Fc-домен дикого типа, индуцируя в результате сигнал с длиной волны от 520 до 620 нм. Полипептидный комплекс, содержащий немеченый мутантный Fc-домен, конкурирует с полипептидным комплексом, содержащим Fc-домен дикого типа, за взаимодействие с Fcγ-рецептором. Относительную аффинность связывания можно оценивать, определяя количественно снижение флуоресценции в результате конкуренции. Методы биотинилирования полипептидных комплексов, таких как антитела, с помощью сульфо-NHS-биотина или подобных агентов являются известными. Приемлемые методы введения GST-метки в Fcγ-рецептор включают методы, которые предусматривают слияние полипептидов, кодирующих Fey и GST, в рамке считывания, экспрессию литого гена с использованием клеток, в которые интродуцирован вектор, несущий ген, и последующую очистку с помощью содержащей глутатион колонки. Индуцированный сигнал предпочтительно можно анализировать, например, посредством подгонки к односайтовой модели конкуренции на основе нелинейного регрессионного анализа с использованием такой программы, как GRAPHPAD PRISM (фирма GraphPad; Сан-Диего).
Одну из субстанций, предназначенных для исследования их взаимодействия, иммобилизуют в качестве лиганда на тонком слое золота сенсорного чипа. Когда свет проникает на заднюю поверхность сенсорного чипа так, что имеет место полное отражение на границе раздела между тонким слоем золота и стеклом, то интенсивность отраженного света в определенном сайте частично снижается (SPR-сигнал). Другую субстанцию, предназначенную для исследования ее взаимодействия, инъецируют в качестве анализируемого вещества на поверхность сенсорного чипа. Когда анализируемое вещество связывается с лигандом, то масса иммобилизованной молекулы-лиганда возрастает.Это изменяет показатель преломления растворителя на поверхности сенсорного чипа. Изменение показателя преломления вызывает положительный сдвиг SPR-сигнала (и наоборот, диссоциация сдвигает сигнал назад в исходное положение). В Biacore-системе уровень описанного выше сдвига (т.е. изменение массы на поверхности сенсорного чипа) откладывают по вертикальной оси, и таким образом в качестве количественных данных получают график изменения массы в зависимости от времени (сенсограмма). Кинетические параметры (константа скорости ассоциации (ка) и константа скорости диссоциации (kd)) определяют из представленных в виде кривых сенсограмм, и определяют аффинность (KD) как соотношение указанных двух констант.BIACORE-методы предпочтительно применяют для анализа ингибирования. Примеры такого анализа ингибирования описаны в Proc. Natl. Acad. Sci. USA 103(11), 2006, cc. 4005-4010.
В контексте настоящего описания «пониженная активность связывания с Fcγ-рецептором» означает, например, что при использовании описанного выше метода анализа конкурентная активность тестируемого полипептидного комплекса составляет 50% или менее, предпочтительно 45% или менее, 40% или менее, 35% или менее, 30% или менее, 20% или менее или 15% или менее и наиболее предпочтительно 10% или менее, 9% или менее, 8% или менее, 7% или менее, 6% или менее, 5% или менее, 4% или менее, 3% или менее, 2% или менее или 1% менее, по сравнению с конкурентной активностью контрольного полипептидного комплекса.
Полипептидные комплексы, содержащие Fc-домен моноклонального антитела в виде IgG1, IgG2, IgG3 или IgG4, можно использовать соответственно в качестве контрольных полипептидных комплексов. Структуры Fc-домена представлены в SEQ ID NO: 23 (А добавлен к N-концу последовательности, представленной в RefSeq под регистрационным номером ААС82527.1), SEQ ID NO: 24 (А добавлен к N-концу последовательности, представленной в RefSeq под регистрационным номером ААВ59393.1), SEQ ID NO: 25 (А добавлен к N-концу последовательности, представленной в RefSeq под регистрационным номером САА27268.1) и SEQ ID NO: 26 (А добавлен к N-концу последовательности, представленной в RefSeq под регистрационным номером ААВ59394.1). Кроме того, когда полипептидный комплекс, содержащий мутантный Fc-домен антитела конкретного изотипа, используют в качестве тестируемой субстанции, то воздействие мутации мутанта на активность связывания с Fcγ-рецептором оценивают с использованием в качестве контроля полипептидного комплекса, содержащего Fc-домен такого же изотипа. Как описано выше, соответственно получают полипептидные комплексы, содержащие мутантный Fc-домен с действительно пониженной активностью связывания с Fcγ-рецептором.
Такие известные мутанты включают, например, мутанты с делецией аминокислот 231A-238S (EU-нумерация) (WO 2009/011941), а также мутанты C226S, C229S, P238S, (C220S) (J. Rheumatol 34, 2007, с. 11); C226S и C229S (Hum. Antibod. Hybridomas 1(1), 1990, cc. 47-54); C226S, C229S, E233P, L234V, и L235A (Blood 109, 2007, cc. 1185-1192).
Предпочтительными полипептидными комплексами являются комплексы, которые содержат Fc-домен с заменой аминокислоты в положении 220, 226, 229, 231, 232, 233, 234, 235, 236, 237, 238, 239, 240, 264, 265, 266, 267, 269, 270, 295, 296, 297, 298, 299, 300, 325, 327, 328, 329, 330, 331 или 332 (EU-нумерация) в аминокислотах, образующих Fc-домен антитела конкретного изотипа. Изобретение не ограничено изотипом антитела, из которого получают Fc-домен, и можно использовать соответствующий Fc-домен, выведенный из моноклонального антитела в виде IgG1, IgG2, IgG3 или IgG4. Предпочтительно следует применять Fc-домен, выведенный из антител в виде IgG1.
Предпочтительными полипептидными комплексами являются, например, комплексы, которые содержат Fc-домен, содержащий одну из указанных ниже замен, положение которой определяется согласно EU-нумерации (каждый номер обозначает положение аминокислотного остатка согласно EU-нумерации; и однобуквенный символ аминокислоты, который находится перед номером, обозначает аминокислотный остаток до его замены, а однобуквенный символ аминокислоты, расположенный за номером, обозначает аминокислотный остаток после замены), аминокислот, образующих Fc-домен антитела в виде IgG1:
(а) L234F, L235E, P331S;
(б) C226S, C229S, P238S;
(в) C226S, C229S или
(г) C226S, C229S, Е233Р, L234V, L235A;
а также комплексы, которые содержат Fc-домен с делецией аминокислот в положениях 231-238.
Кроме того, предпочтительными полипептидными комплексами являются также комплексы, которые содержат Fc-домен, содержащий одну из указанных ниже замен, положение которой определяют согласно EU-нумерации, аминокислот, образующих Fc-домен антитела в виде IgG2:
(д) H268Q, V309L, A330S и P331S;
(е) V234A;
(ж) G237A;
(з) V234A и G237A;
(и) А235Е и G237A или
(к) V234A, А235Е и G237A. Каждый номер обозначает положение аминокислотного остатка согласно EU-нумерации; и однобуквенный символ аминокислоты, который находится перед номером, обозначает аминокислотный остаток до его замены, а однобуквенный символ аминокислоты, расположенный за номером, обозначает аминокислотный остаток после замены.
Кроме того, предпочтительными полипептидными комплексами являются также комплексы, которые содержат Fc-домен, содержащий одну из указанных ниже замен, положение которой определяют согласно EU-нумерации, аминокислот, образующих Fc-домен антитела в виде IgG3:
(л) F241A;
(м) D265A или
(н) V264A. Каждый номер обозначает положение аминокислотного остатка согласно EU-нумерации; и однобуквенный символ аминокислоты, который находится перед номером, обозначает аминокислотный остаток до его замены, а однобуквенный символ аминокислоты, расположенный за номером, обозначает аминокислотный остаток после замены.
Кроме того, предпочтительными полипептидными комплексами являются также комплексы, которые содержат Fc-домен, содержащий одну из указанных ниже замен, положение которой определяют согласно EU-нумерации, аминокислот, образующих Fc-домен антитела в виде IgG4:
(о) L235A, G237A и Е318А;
(п) L235E или
(р) F234A и L235A. Каждый номер обозначает положение аминокислотного остатка согласно EU-нумерации; и однобуквенный символ аминокислоты, который находится перед номером, обозначает аминокислотный остаток до его замены, а однобуквенный символ аминокислоты, расположенный за номером, обозначает аминокислотный остаток после замены.
Другие предпочтительные полипептидные комплексы представляют собой комплексы, содержащие Fc-домен, в котором любая аминокислота в положении 233, 234, 235, 236, 237, 327, 330 или 331 (EU-нумерация) в аминокислотах, образующих Fc-домен антитела в виде IgG1, заменена на аминокислоту, которая находится в соответствующем положении согласно EU-нумерации, в соответствующем IgG2 или IgG4.
Предпочтительные полипептидные комплексы представляют собой также комплексы, содержащие Fc-домен, в котором одна или несколько аминокислот в положениях 234, 235 и 297 (EU-нумерация) в аминокислотах, образующих Fc-домен антитела в виде IgG1, заменена(ы) на другие аминокислоты. Изобретение не ограничено типом аминокислоты после замены; однако наиболее предпочтительными являются полипептидные комплексы, содержащие Fc-домен, в котором одна или несколько аминокислот в положениях 234, 235 и 297 заменена(ы) на аланин.
Предпочтительные полипептидные комплексы представляют собой также комплексы, содержащие Fc-домен, в котором аминокислота в положении 265 (EU-нумерация) в аминокислотах, образующих Fc-домен антитела в виде IgG1, замена другой аминокислотой. Изобретение не ограничено типом аминокислоты после замены; однако наиболее предпочтительными являются полипептидные комплексы, содержащие Fc-домен, в котором аминокислота в положении 265 заменена на аланин.
Fc-домен, выведенный из биспецифического антитела
Согласно настоящему описанию соответствующие Fc-домены, выведенные из биспецифического антитела, можно применять также в качестве Fc-домена с пониженной активностью связывания Fcγ-рецептора. Биспецифическое антитело представляет собой антитело, которое имеет две различные специфичности. Биспецифическое антитело IgG-типа можно секретировать из гибридной гибридомы (квадромы), полученной путем слияния двух типов гибридом, продуцирующих антитело в виде IgG (Milstein С. и др., Nature 305, 1983, сс. 537-540).
Альтернативно этому, биспецифическое антитело IgG-типа можно секретировать также посредством интродукции в клетки в целом четырех генов, т.е. генов L-цепей и Н-цепей, образующих представляющие интерес IgG двух типов, и их коэкспрессии. Однако теоретически существует десять комбинаций Н-цепей и L-цепей IgG, которые можно получать такими методами. Трудно очищать IgG, состоящий из требуемой комбинации Н-цепей и L-цепей из десяти типов IgG. Кроме того, теоретическое количество секретируемого IgG с требуемой комбинацией, также в значительной степени снижено, поэтому требуется осуществлять крупномасштабное культивирование. Это дополнительно увеличивает стоимость производства.
В таком случае соответствующую аминокислотную замену можно интродуцировать в СН3-домен, образующей Fc-домен Н-цепи, для того, чтобы предпочтительно секретировать IgG с гетерологичной комбинацией Н-цепей. В частности, этот метод можно осуществлять путем замены аминокислоты, которая имеет более крупную боковую цепь (выступ (что означает «выпуклость»)), на аминокислоту в СН3-домене одной из Н-цепей, и замены аминокислоты, которая имеет менее крупную боковую цепи (впадина (что означает «пустота»)), на аминокислоту в СН3-домене другой Н-цепи так, что выступ помещается во впадину. Это способствует образованию гетеромерной Н-цепи и одновременно ингибирует образование гомомерной Н-цепи (WO 1996/027011; Ridgway J.B. и др., Protein Engineering 9, 1996, сс.617-621; Merchant A.M. и др., Nature Biotechnology 16, 1998, сс.677-681).
С другой стороны, касательно L-цепи, вариабельная область L-цепи является менее полиморфной, чем вариабельная область Н-цепи, и следовательно можно ожидать получения общей L-цепи, которая может обладать способностью связываться с обоими Н-цепями. Для достижения эффективной экспрессии биспецифического IgG можно применять интродукцию генов такой общей L-цепи и двух Н-цепей в клетки, которые экспрессируют IgG (Nature Biotechnology 16, 1998, сс.677-681). Однако эту идею трудно реализовать, поскольку вероятность получения путем случайного отбора двух типов антител, содержащих одинаковую L-цепь, является низкой. Таким образом, предложен метод селекции общей L-цепи, для которой характерна выраженная способностью к связыванию с любыми различными Н-цепями (WO 2004/065611).
Кроме того, существуют также известные методики получения биспецифического антитела путем применения методов контроля полипептидной ассоциации или ассоциации образованных из полипептидов гетеромерных мультимеров к ассоциации между двумя полипептидами, которые образуют Fc-домен. В частности, методы контроля полипептидной ассоциации можно применять для получения биспецифического антитела (WO 2006/106905), в котором аминокислотные остатки, образующие поверхность раздела между двумя полипептидами, которые образуют Fc-домен, изменены для ингибирования ассоциации между Fc-доменами, имеющими одинаковую последовательность, и обеспечения образования полипептидных комплексов, образованных двумя Fc-доменами, имеющими различные последовательности.
Два указанных выше полипептида, которые образуют Fc-домен, выведенный из биспецифического антитела, можно соответственно применять в качестве домена, включающего в себя Fc-домен, предлагаемый в настоящем изобретении. Более конкретно, указанные два предпочтительных полипептида, которые образуют Fc-домен, включают полипептиды, в которых аминокислоты в положениях 349 и 366 (EU-нумерация) в аминокислотной последовательности одного полипептида представляют собой цистеин и триптофан соответственно, а аминокислоты в положениях 356, 366, 368 и 407 (EU-нумерация) в аминокислотной последовательности другого полипептида представляют собой цистеин, серии, аланин и валин соответственно.
В другом варианте осуществления изобретения предпочтительные домены, включающие в себя Fc-домен, предлагаемый в настоящем изобретении, включают два полипептида, которые образуют Fc-домен, в котором аминокислота в положении 409 (EU-нумерация) в аминокислотной последовательности одного полипептида представляет собой аспарагиновую кислоту, а аминокислота в положении 399 (EU-нумерация) в аминокислотной последовательности другого полипептида представляет собой лизин. Согласно указанному выше варианту осуществления изобретения аминокислота в положении 409 может представлять собой глутаминовую кислоту вместо аспарагиновой кислоты, а аминокислота в положении 399 может представлять собой аргинин вместо лизина. Кроме того, аспарагиновую кислоту в положении 360 или 392 предпочтительно можно также применять в сочетании с лизином в положении 399.
В другом варианте осуществления изобретения предпочтительные домены, включающие в себя Fc-домен, предлагаемый в настоящем изобретении, включают два полипептида, которые образуют Fc-домен, в котором аминокислота в положении 370 (EU-нумерация) в аминокислотной последовательности одного полипептида представляет собой глутаминовую кислоту, а аминокислота в положении 357 (EU-нумерация) в аминокислотной последовательности другого полипептида представляет собой лизин.
В следующем варианте осуществления изобретения предпочтительные домены, включающие в себя Fc-домен, предлагаемый в настоящем изобретении, включают два полипептида, которые образуют Fc-домен, в котором аминокислота в положении 439 (EU-нумерация) в аминокислотной последовательности одного полипептида представляет собой глутаминовую кислоту, а аминокислота в положении 356 (EU-нумерация) в аминокислотной последовательности другого полипептида представляет собой лизин.
Кроме того, предпочтительные домены, включающие в себя Fc-домен, предлагаемый в настоящем изобретении, включают варианты с их комбинацией:
два полипептида образуют Fc-домен, в котором аминокислоты в положениях 409 и 370 (EU-нумерация) в аминокислотной последовательности одного полипептида представляют собой аспарагиновую кислоту и глутаминовую кислоту соответственно, а аминокислоты в положениях 399 и 357 (EU-нумерация) в аминокислотной последовательности другого полипептида обе представляют собой лизин (в этом варианте осуществления изобретения глутаминовую кислоту в положении 370 можно заменять на аспарагиновую кислоту, и глутаминовую кислоту в положении 370 можно заменять на аспарагиновую кислоту в положении 392);
два полипептида образуют Fc-домен, в котором аминокислоты в положениях 409 и 439 (EU-нумерация) в аминокислотной последовательности одного полипептида представляют собой аспарагиновую кислоту и глутаминовую кислоту соответственно, а аминокислоты в положениях 399 и 356 (EU-нумерация) в аминокислотной последовательности другого полипептида обе представляют собой лизин (в этом варианте осуществления изобретения глутаминовую кислоту в положении 439 можно заменять на аспарагиновую кислоту в положении 360, 392 или 439);
два полипептида образуют Fc-домен, в котором аминокислоты в положениях 370 и 439 (EU-нумерация) в аминокислотной последовательности одного полипептида обе представляют собой глутаминовую кислоту, а аминокислоты в положениях 357 и 356 (EU-нумерация) в аминокислотной последовательности другого полипептида обе представляют собой лизин; и
два полипептида образуют Fc-домен, в котором аминокислоты в положениях 409,370 и 439 (EU-нумерация) в аминокислотной последовательности одного полипептида представляют собой аспарагиновую кислоту, глутаминовую кислоту и глутаминовую кислоту соответственно, а аминокислоты в положениях 399, 357 и 356 (EU-нумерация) в аминокислотной последовательности другого полипептида все представляют собой лизин (в этом варианте осуществления изобретения аминокислоту в положении 370 нельзя заменять на глутаминовую кислоту; альтернативно этому, вместо замены аминокислоты в положении 370 на глутаминовую кислоту глутаминовую кислоту в положении 439 можно заменять на аспарагиновую кислоту или глутаминовую кислоту в положении 439 можно заменять на аспарагиновую кислоту в положении 392).
В следующем варианте осуществления изобретения предпочтительные домены, включающие в себя Fc-домен, предлагаемый в настоящем изобретении, включают два полипептида, которые образуют Fc-домен, в котором аминокислота в положении 356 (EU-нумерация) в аминокислотной последовательности одного полипептида представляет собой лизин, а аминокислоты в положениях 435 и 439 (EU-нумерация) в аминокислотной последовательности другого полипептида представляет собой аргинин и глутаминовую кислоту соответственно.
Когда два указанных выше полипептида, которые образуют Fc-домен, выведенный из биспецифического антитела, применяют в качестве домена, включающего в себя Fc-домен, предлагаемый в настоящем изобретении, то антигенсвязывающие домены и/или CD3-связывающие домены, предлагаемые в настоящем изобретении, можно располагать в требуемой комбинации.
Fc-домен с пониженной С-концевой гетерогенностью
Согласно настоящему описанию Fc-домены, отличающиеся улучшенной С-концевой гетерогенностью в дополнение к указанным выше характеристикам, можно применять в качестве Fc-доменов с пониженной активностью связывания Fcγ-рецептора. Более конкретно, в настоящем изобретении предложены Fc-домены, в которых глицин и лизин в положениях 446 и 447 (EU-нумерация) соответственно в аминокислотных последовательностях двух полипептидов, образующих Fc-домен, который выведен из IgG1, IgG2, IgG3 или IgG4, удаляют путем делеции.
Домен, связывающий комплекс Т-клеточного рецептора
В контексте настоящего описания «домен, связывающий комплекс Т-клеточного рецептора» относится участку антитела к комплексу Т-клеточного рецептора, который содержит область, которая специфически связывается и является комплементарной всему комплексу Т-клеточного рецептора или его части. Указанный комплекс Т-клеточного рецептора может представлять собой сам Т-клеточный рецептор или молекулу-адаптер, которая в сочетании с Т-клеточным рецептором образует комплекс Т-клеточного рецептора. Предпочтительным адаптером является CD3.
Домен, связывающий Т-клеточный рецептор
В контексте настоящего описания «домен, связывающий Т-клеточный рецептор» относится участку антитела к Т-клеточному рецептору, который содержит область, которая специфически связывается и является комплементарной всему Т-клеточному рецептору или его части.
Можно применять вариабельную область или константную область Т-клеточного рецептора. Однако предпочтительными эпитопами, с которыми связывается CD3-связывающий домен, являются эпитопы, локализованные в константной области. Последовательности константной области включают, например, последовательности α- цепи Т-клеточного рецептора (RefSeq, регистрационный номер САА26636.1; SEQ ID NO: 67), р-цепи Т-клеточного рецептора (RefSeq, регистрационный номер accession number С25777; SEQ ID NO: 68), yl-цепи Т-клеточного рецептора (RefSeq, регистрационный номер А26659; SEQ ID NO: 69), у2-цепи Т-клеточного рецептора (RefSeq, регистрационный номер ААВ63312.1; SEQ ID NO: 70) и 6 -цепи Т-клеточного рецептора (RefSeq, регистрационный номер ААА61033.1; SEQ ID NO: 71).
CD3-связывающий домен
В контексте настоящего описания «CD3-связывающий домен» относится к участку антитела к CD3, содержащему область, которая специфически связывается и является комплементарной всему CD3 или его части. CD3-связывающий домен можно выводить из одного или нескольких вариабельных доменов антитела. Предпочтительно CD3-связывающий домен включает как вариабельную область легкой цепи (VL) антитела к CD3, так и вариабельную область тяжелой цепи (VH) антитела к CD3. Указанные предпочтительные CD3-связывающие домены включают, например, «одноцепочечный Fv (scFv)», «одноцепочечное антитело», «Fv», «одноцепочечный Fv2 (scFv2)», «Fab» и «F(ab')2».
CD3-связывающий домен, предлагаемый в настоящем изобретении, может связываться с любым эпитопом, если эпитоп локализован внутри у-цепи, 5-цепи или s-цепи последовательности, образующей человеческий CD3. Согласно настоящему изобретению предпочтительные CD3-связывающие домены включают домены, которые содержат вариабельную область легкой цепи (VL) антитела к CD3 и вариабельную область тяжелой цепи (VH) антитела к CD3, которые связываются с эпитопом, который находится во внеклеточном домене s-цепи человеческого комплекса CD3. Указанные предпочтительные CD3-связывающие домены включают домены, которые содержат вариабельную область легкой цепи (VL) антитела к CD3 и вариабельную область тяжелой цепи (VH) антитела ОКТЗ (Proc. Natl. Acad. Sci. USA 77, 1980, cc. 4914-4917) или различных известных антител к CD3. Кроме того, указанные приемлемые CD3-связывающие домены включают домены, выведенные из антитела к CD3 с требуемыми характеристиками, которые получают путем иммунизации требуемого животного γ-цепью, δ-цепью или ε-цепью, образующей человеческий CD3, с использованием описанных выше методов. Приемлемыми антителами к CD3, из которых выводят CD3-связывающий домен, являются человеческие антитела и антитела, гуманизированные соответствующим описанным выше методом. Структуры γ-цепи, δ-цепи и ε-цепи, которые образуют CD3, представлены в виде полинуклеотидных последовательностей SEQ ID NO: 27 (NM 000073.2), SEQ ID NO: 29 (NM 000732.4) и SEQ ID NO: 31 (NM 000733.3) соответственно и в виде полипептидных последовательностей SEQ ID NO: 28 (NP 000064.1), SEQ ID NO: 30 (NP 000723.1) и SEQ ID NO: 32 (NP 000724.1) соответственно (в скобках представлены регистрационные номера RefSeq).
Полипептидный комплекс
На структуру полипептидного комплекса, предлагаемого в настоящем изобретении, не накладываются ограничения, если он содержит
(1) антигенсвязывающий домен;
(2) домен, содержащий Fc-домен с пониженной активностью связывания с Fcγ-рецептором; и
(3) домен, связывающий комплекс Т-клеточного рецептора, указанный выше.
Согласно настоящему изобретению предпочтительным доменом, связывающим комплекс Т-клеточного рецептора, является домен, связывающий Т-клеточный рецептор, или CD3-связывающий домен. Каждый из описанных выше доменов может быть сцеплен непосредственно через пептидную связь. Например, (1) F(ab')2 применяют в качестве антигенсвязывающего домена и (2) Fc-домен с пониженной активностью связывания Fcγ-рецептора применяют в качестве домена, который содержит Fc-домен с пониженной активностью связывания с Fcγ-рецептором. В этом случае, когда антигенсвязывающий домен (1) соединяют через пептидную связь с доменом, который содержит Fc-домен (2), то сцепленный полипептид образует структуру антитела. Указанное антитело можно получать путем очистки культуральных сред описанных выше гибридом или очистки культуральных сред требуемых клеток-хозяев, стабильно несущих полинуклеотид, который кодирует образующий антитело полипептид.
Когда CD3-связывающий домен (3) соединяют со структурой антитела, то CD3-связывающий домен можно соединять через пептидную связь с С-концом константной области структуры антитела. В другом варианте осуществления изобретения CD3-связывающий домен соединяют через пептидную связь с N-концом вариабельной области тяжелой цепи или вариабельной области легкой цепи структуры антитела. В другом варианте осуществления изобретения CD3-связывающий домен можно соединять через пептидную связь с С-концом константной области легкой цепи структуры антитела. Подлежащий соединению CD3-связывающий домен может иметь любую требуемую структуру; однако указанный приемлемый CD3-связывающий домен включает предпочтительно Fv и более предпочтительно scFv. На валентность CD3-связывающего домена, который связывается со структурой антитела, ограничения не накладываются. Для соединения двухвалентного CD3-связывающего домена со структурой антитела одновалентный CD3-связывающий домен можно соединять через пептидную связь с соответствующими С-концами двух Fc-доменов, образующих константную область структуры антитела. Альтернативно этому, для соединения двухвалентного CD3-связывающего домена со структурой антитела двухвалентный scFv (т.е. sc(Fv)2) можно соединять через пептидную связь с С-концом одного из двух Fc-доменов. В этом случае можно успешно получать полипептидный комплекс, в котором двухвалентный scFv (т.е. sc(Fv)2) связан с С-концом только одного из двух Fc-доменов, образующих константную область структуры антитела, с использованием указанного выше Fc-домена, выведенного из биспецифического антитела. Альтернативно этому, для соединения одновалентного CD3-связывающего домена со структурой антитела одновалентный scFv можно соединять через пептидную связь с С-концом одного из двух Fc-доменов. В этом случае можно успешно получать полипептидный комплекс, предлагаемый в настоящем изобретении, в котором одновалентный scFv сцеплен с С-концом только одного из двух Fc-доменов, образующих константную область структуры антитела, используя указанный выше Fc-домен, выведенный из биспецифического антитела.
Кроме того, когда CD3-связывающий домен (3) соединяют через пептидную связь с С-концом константной области структуры антитела, то приемлемые полипептидные комплексы включают комплексы, в которых Fv тяжелой цепи, образующий CD3-связывающий домен, соединяют с С-концом одной константной области (СН3-домен), образующей Fc-домен, и Fv-фрагмент легкой цепи, образующий CD3-связывающий домен, соединяют с С-концом другой константной области (СН3-домен), образующей Fc-домен. В этом случае для соединения Fv-фрагмента тяжелой цепи или легкой цепи с С-концом константной области (СН3-домен) встраивают приемлемый линкер, такой как Gly-Gly-Gly-Gly-Ser (SEQ ID NO: 7). Количество повторов в линкере не имеет решающего значения; однако их можно выбирать из 1-10, предпочтительно 2-8 или более предпочтительно 2-6. В частности, можно встраивать приемлемый линкер, в котором количество повторов [Gly-Gly-Gly-Gly-Ser] (SEQ ID NO: 7) составляет 1, 2, 3, 4, 5, 6, 7, 8, 9 или 10.
Альтернативно этому, когда получают полипептидный комплекс, в котором Fv-фрагмент тяжелой цепи, образующий CD3-связывающий домен, соединен с С-концом одной константной области (СН3-домен), образующей Fc-домен, и Fv-фрагмент легкой цепи CD3-связывающего домена соединен с С-концом другой константной области (СН3-домен), образующей Fc-домен, то для повышения ассоциации между Fv-фрагментом тяжелой цепи и Fv-фрагментом легкой цепи можно осуществлять соответствующие изменения аминокислотных остатков, которые позволяют образовывать дисульфидные мостики между Fv-фрагментом тяжелой цепи и Fv-фрагментом легкой цепи.
В другом варианте осуществления изобретения, когда получают полипептидный комплекс, предлагаемый в настоящем изобретении, в котором Fv-фрагмент тяжелой цепи, образующий CD3-связывающий домен, соединен с С-концом одной константной области (СН3-домен), образующей Fc-домен, а Fv-фрагмент легкой цепи, образующий CD3-связывающий домен, соединен С-концом другой константной области (СН3-домен), образующей Fc-домен, то для повышения ассоциации между Fv-фрагментом тяжелой цепи и Fv-фрагментом легкой цепи СН1- и CL-домены можно соединять и с Fv-фрагментом тяжелой цепи и с Fv-фрагментом легкой цепи.
Согласно еще одному варианту осуществления изобретения для того, чтобы соединять двухвалентный CD3-связывающий домен со структурой антитела, одновалентный CD3-связывающий домен можно соединять через пептидную связь с соответствующими С-концами двух константных областей легких цепей или соответствующими N-концами вариабельных областей легких цепей структуры антитела. Альтернативно этому, для того, чтобы соединять двухвалентный CD3-связывающий домен со структурой антитела, двухвалентный scFv (т.е. sc(Fv)2) можно соединять через пептидную связь с соответствующими С-концами двух константных областей легких цепей или соответствующими N-концами вариабельных областей легких цепей структуры антитела. В этом случае, используя описанный выше Fc-домен, выведенный из биспецифического антитела, можно эффективно получать полипептидные комплексы, в которых двухвалентный scFv (т.е. sc(Fv)2) связан с С- или N-концом одной из двух вариабельных областей легких цепей структуры антитела. Альтернативно этому, для того, чтобы соединять одновалентный CD3-связывающий домен со структурой антитела, одновалентный scFv можно соединять через пептидную связь с С- или N-концом одной из двух вариабельных областей легких цепей. В этом случае, используя описанный выше Fc-домен, выведенный из биспецифического антитела, можно эффективно получать полипептидные комплексы, в которых одновалентный scFv связан с N-или С-концом одной вариабельной области легкой цепи из двух вариабельных областей легких цепей структуры антитела.
В другом варианте осуществления изобретения для того, чтобы соединять двухвалентный CD3-связывающий домен со структурой антитела, одновалентный CD3-связывающий домен можно соединять через пептидную связь с соответствующими N концами двух вариабельных областей тяжелых цепей структуры антитела. Альтернативно этому, для того, чтобы соединять двухвалентный CD3-связывающий домен со структурой антитела, двухвалентный scFv (т.е. sc(Fv)2) можно соединять через пептидную связь с N-концом одной из двух вариабельных областей тяжелых цепей. В этом случае, используя описанный выше Fc-домен, выведенный из биспецифического антитела, можно эффективно получать полипептидные комплексы, в которых двухвалентный scFv (т.е. sc(Fv)2) связан с N-концом только одной из двух вариабельных областей тяжелых цепей структуры антитела. Альтернативно этому, для того, чтобы соединять одновалентный CD3-связывающий домен со структурой антитела, одновалентный scFv можно соединять через пептидную связь с N-концом одной из двух вариабельных областей тяжелых цепей. В этом случае, используя описанный выше Fc-домен, выведенный из биспецифического антитела, можно эффективно получать полипептидные комплексы, в которых одновалентный scFv связан с N-концом одной из двух вариабельных областей тяжелых цепей структуры антитела.
Кроме того, описанный выше полипептидный комплекс можно получать путем соединения каждого домена непосредственно через пептидную связь или посредством пептидного связывания через пептидный линкер. В этом случае в качестве линкера, который следует применять, можно использовать, например, описанный выше линкер и соответствующие линкеры с пептидной меткой, например, с His-меткой, НА-меткой или FLAG-меткой. Кроме того, предпочтительно можно использовать способность к взаимному связыванию на основе водородной связи, дисульфидной связи, ковалентной связи или ионного взаимодействия, или их комбинации. Например, можно использовать аффинность между СН1 и CL антитела или можно применять описанные выше Fc-домены, выведенные из биспецифического антитела, для гетеромерной ассоциации Fc-доменов. Кроме того, предпочтительно можно использовать внутридоменные дисульфидные мостики, как это описано в примерах.
В другой структуре полипептидного комплекса, предлагаемого в настоящем изобретении, например, одновалентный Fv и одновалентный Fab предпочтительно применяют в качестве (1) антигенсвязывающего домена. В этом случае применяют представленную ниже структуру. Fv-фрагмент тяжелой цепи (VH) или Fv-фрагмент легкой цепи (VL) одновалентного Fv соединяют через пептидную связь с CH1-доменом тяжелой цепи. CH1-домен тяжелой цепи соединяют через пептидную связь с одним из (2) двух Fc-доменов с пониженной активностью связывания с Fcγ-рецептором, которые образуют полипептидный комплекс, предлагаемый в настоящем изобретении. Другой VL- или VH-фрагмент одновалентного Fv соединяют через пептидную связь с CL-доменом легкой цепи, который соединен через дисульфидный мостик с СН1-доменом тяжелой цепи. Таким образом, VH и VL соответственно соединенные с концами CH1-домена тяжелой цепи и CL-домена легкой цепи, образуют связывающий домен антитела. sc(Fv)2, который образует как (1) связывающий домен антитела, так и (3) CD3-связывающий домен, можно соединять через пептидную связь с N-концом другого Fc-домена из двух описанных выше доменов. В этом случае полипептидный комплекс, который имеет структуру, в которой СН1-домен тяжелой цепи соединен через пептидную связь с одним из двух Fc-доменов, образующих полипептидный комплекс, и sc(Fv)2 соединен через пептидную связь с другим Fc-доменом, можно получать, используя описанный выше Fc-домен, выведенный из биспецифического антитела. Описанный выше полипептидный комплекс можно получать путем соединения каждого домена непосредственно через пептидную связь или путем пептидного связывания через пептидный линкер. В этом случае линкер, который следует применять, представляет собой, например, описанные выше в качестве примера линкеры и соответствующие линкеры с пептидной меткой, например, с His-меткой, НА-меткой или FLAG-меткой.
В другой предпочтительной структуре полипептидного комплекса, предлагаемого в настоящем изобретении, в качестве (1) антигенсвязывающего домена применяют, например, также двухвалентный scFv. В одном из вариантов структуры можно получать также полипептидный комплекс, в котором один из двухвалентных scFv соединяют через пептидную связь посредством VH, образующей (3) CD3-связывающий домен, с одним из двух (2) Fc-доменов с пониженной активностью связывания с Fcγ-рецептором, а другой двухвалентный scFv соединяют через пептидную связь посредством VL, образующей (3) CD3-связывающий домен, с одним из двух (2) Fc-доменов с пониженной активностью связывания с Fcγ-рецептором. В этом случае можно применять описанный выше Fc-домен, выведенный из биспецифического антитела. Описанный выше полипептидный комплекс можно получать путем соединения каждого домена непосредственно через пептидную связь или путем пептидного связывания через пептидный линкер. В этом случае линкер, который следует применять, представляет собой, например, описанные выше в качестве примера линкеры и соответствующие линкеры с пептидной меткой, например, с His-меткой, НА-меткой или FLAG-меткой.
В другом варианте структуры, для создания которой используют двухвалентный scFv в качестве (1) антигенсвязывающего домена, можно получать полипептидный комплекс, в котором один из двухвалентных scFv соединен через пептидную связь scFv, образующего (3) CD3-связывающий домен, с одним из двух (2) Fc-доменов с пониженной активностью связывания с Fcγ-рецептором, а другой двухвалентный scFv соединен через пептидную связь с другим (2) Fc-доменом с пониженной активностью связывания с Fcγ-рецептором. В этом случае, используя описанный выше Fc-домен, выведенный из биспецифического антитела, можно получать полипептидный комплекс, в котором scFv, образующий антигенсвязывающий домен, соединен через пептидную связь посредством scFv, образующего CD3-связывающий домен, с одним из двух Fc-доменов, образующих полипептидный комплекс, и scFv, образующий антигенсвязывающий домен, соединен через пептидную связь с другим Fc-доменом. Описанный выше полипептидный комплекс можно получать путем соединения каждого домена непосредственно через пептидную связь или путем пептидного связывания через пептидный линкер. В этом случае линкер, который можно применять, представляет собой, например, описанные выше в качестве примера линкеры и соответствующие линкеры с пептидной меткой, например, с His-меткой, НА-меткой или FLAG-меткой.
В другой предпочтительной структуре полипептидного комплекса, предлагаемого в настоящем изобретении, например, как антигенсвязывающий домен, так и домен, связывающий комплекс Т-клеточного рецептора, каждый присутствует в структуре одновалентного Fab. Для создания одного из вариантов структуры можно получать полипептидный комплекс, в котором Fv-фрагмент тяжелой цепи одновалентного Fab, образующего антигенсвязывающий домен, соединен посредством CH1-домена с одним из полипептидов, образующих Fc-домен, и Fv-фрагмент легкой цепи Fab соединен с CL-доменом, в то время как Fv-фрагмент тяжелой цепи Fab, образующего связывающий Т-клеточный рецептор домен соединен посредством CH1-домена с другим полипептидом, образующим Fc-домен, а Fv-фрагмент легкой цепи Fab соединен с CL-доменом.
Для создания другого варианта структуры можно получать полипептидный комплекс, в котором Fv-фрагмент тяжелой цепи одновалентного Fab, образующего антигенсвязывающий домен, соединен посредством СН1-доменас одним полипептидом, образующим Fc-домен, и Fv-фрагмент легкой цепи Fab соединен с CL-доменом, а Fv-фрагмент легкой цепи Fab, образующего домен, связывающий Т-клеточный рецептор, соединен посредством CH1-домена с другим полипептидом, образующим Fc-домен, и Fv-фрагмент тяжелой цепи Fab соединен с CL-доменом. Альтернативно этому, можно получать также полипептидный комплекс, в котором Fv-фрагмент тяжелой цепи одновалентного Fab, образующего домен, связывающий Т-клеточный рецептор, соединен посредством CH1-домена с одним полипептидом, образующим Fc-домен, и Fv-фрагмент легкой цепи Fab соединен с CL-доменом, Fv-фрагмент легкой цепи Fab, образующего антигенсвязывающий домен, соединен посредством CH1-домена с другим полипептидом, образующим Fc-домен, и Fv-фрагмент тяжелой цепи Fab соединен с CL-доменом.
Для создания другого варианта структуры можно получать полипептидный комплекс, в котором Fv-фрагмент тяжелой цепи одновалентного Fab, образующего антигенсвязывающий домен, соединен посредством CH1-домена с одним полипептидом, образующим Fc-домен, и Fv-фрагмент легкой цепи Fab соединен с CL-доменом, а Fv-фрагмент легкой цепи Fab, образующего домен, связывающий Т-клеточный рецептор, соединен посредством CL-домена с другим полипептидом, образующим Fc-домен, и Fv-фрагмент легкой цепи Fab соединен с СН1-доменом. Альтернативно этому, можно получать также полипептидный комплекс, в котором Fv-фрагмент тяжелой цепи одновалентного Fab, образующего домен, связывающий Т-клеточный рецептор, соединен посредством CH1-домена с одним полипептидом, образующим Fc-домен, и Fv-фрагмент легкой цепи Fab соединен с CL-доменом, а Fv-фрагмент тяжелой цепи Fab, образующего антигенсвязывающий домен, соединен посредством CL-домена с другим полипептидом, образующим Fc-домен, и Fv-фрагмент легкой цепи Fab соединен с СН1-доменом.
В другом варианте структуры полипептидного комплекса, предлагаемого в настоящем изобретении, в котором как антигенсвязывающий домен, так и домен, связывающий комплекс Т-клеточного рецептора, каждый имеет структуру одновалентного Fab, предпочтительные полипептиды, предлагаемые в настоящем изобретении, включают полипептиды, которые имеют:
(1) антигенсвязывающий домен, в котором Fv-фрагмент тяжелой цепи структуры одновалентного Fab, который связывается с антигеном, соединен посредством CH1-домена одного из описанных выше полипептидов, образующих Fc-домен, и Fv-фрагмент структуры Fab соединен с CL-доменом; и
(2) домен, связывающий комплекс Т-клеточного рецептора, в котором Fv-фрагмент тяжелой цепи структуры одновалентного Fab, который связывается с комплексом Т-клеточного рецептора, соединен посредством CH1-домена с другим полипептидом, образующим Fc-домен, и Fv-фрагмент легкой цепи структуры Fab сцеплен с CL-доменом;
и в которых электрические заряды СН1- и CL-доменов контролируют таким образом, чтобы имела место ассоциация Fv-фрагмента тяжелой цепи антигенсвязывающего домена с Fv-фрагментом легкой цепи антигенсвязывающего домена или ассоциация Fv-фрагмента тяжелой цепи домена, связывающего Т-клеточный рецептор, с Fv-фрагментом легкой цепи домена, связывающего Т-клеточный рецептор. В этом варианте осуществления изобретения структура (структура с контролируемой ассоциацией) полипептидного комплекса не ограничена конкретной структурой, если электрические заряды СН1- и CL-доменов контролируются таким образом, чтобы имела место ассоциация Fv-фрагмента тяжелой цепи антигенсвязывающего домена с Fv-фрагментом легкой цепи антигенсвязывающего домена или ассоциация Fv-фрагмента тяжелой цепи домена, связывающего Т-клеточный рецептор, с Fv-фрагментом легкой цепи домена, связывающего Т-клеточный рецептор.
Для создания варианта структуры с контролируемой ассоциацией можно получать полипептидный комплекс, в котором аминокислотные остатки СН1-домена, сцепленного с Fv-фрагментом тяжелой цепи домена, связывающего комплекс Т-клеточного рецептора, имеют такие же электрические заряды, что и аминокислотные остатки CL-домена, сцепленного с Fv-фрагментом легкой цепи антигенсвязывающего домена.
Для создания варианта структуры с контролируемой ассоциацией можно получать полипептидный комплекс, в котором аминокислотные остатки СН1-домена, сцепленного с Fv-фрагментом тяжелой цепи антигенсвязывающего домена, имеют такие же электрические заряды, что и аминокислотные остатки CL-домена, сцепленного с Fv-фрагментом легкой цепи домена, связывающего комплекс Т-клеточного рецептора.
Для создания варианта структуры с контролируемой ассоциацией можно получать полипептидный комплекс, в котором аминокислотные остатки СН1-домена, сцепленного с Fv-фрагментом тяжелой цепи домена, связывающего комплекс Т-клеточного рецептора, имеют такие же электрические заряды, что и аминокислотные остатки CL-домена, сцепленного с Fv-фрагментом легкой цепи антигенсвязывающего домена, и аминокислотные остатки СН1-домена, сцепленного с Fv-фрагментом тяжелой цепи антигенсвязывающего домена, имеют такие же электрические заряды, что и аминокислотные остатки CL-домена, сцепленного с Fv-фрагментом легкой цепи домена, связывающего комплекс Т-клеточного рецептора.
Для создания другого варианта структуры с контролируемой ассоциацией можно получать полипептидный комплекс, в котором аминокислотные остатки CH1-домена, сцепленного с Fv-фрагментом тяжелой цепи домена, связывающего комплекс Т-клеточного рецептора, имеют такие же электрические заряды, что и аминокислотные остатки CL-домена, сцепленного с Fv-фрагментом легкой цепи антигенсвязывающего домена, и аминокислотные остатки СН1-домена, сцепленного с Fv-фрагментом тяжелой цепи домена, связывающего комплекс Т-клеточного рецептора, имеют электрические заряды, противоположные зарядам аминокислотных остатков CL-домена, сцепленного с Fv-фрагментом легкой цепи домена, связывающего Т-клеточный рецептор.
Для создания еще одного варианта структуры с контролируемой ассоциацией можно получать полипептидный комплекс, в котором аминокислотные остатки CH1-домена, сцепленного с Fv-фрагментом тяжелой цепи домена, связывающего комплекс Т-клеточного рецептора, имеют такие же электрические заряды, что и аминокислотные остатки CL-домена, сцепленного с Fv-фрагментом легкой цепи антигенсвязывающего домена; аминокислотные остатки СН1-домена, сцепленного с Fv-фрагментом тяжелой цепи антигенсвязывающего домена, имеют такие же электрические заряды, что и остатки CL-домена, сцепленного с Fv-фрагментом легкой цепи домена, связывающего комплекс Т-клеточного рецептора; аминокислотные остатки СН1-домена, сцепленного с Fv-фрагментом тяжелой цепи домена, связывающего комплекс Т-клеточного рецептора, имеют электрические заряды, противоположные зарядам аминокислотных остатков CL-домена, сцепленного с Fv-фрагментом легкой цепи домена, связывающего Т-клеточный рецептор.
Для создания еще одного варианта структуры с контролируемой ассоциацией можно получать полипептидный комплекс, в котором аминокислотные остатки CH1-домена, сцепленного с Fv-фрагментом тяжелой цепи антигенсвязывающего домена, имеют такие же электрические заряды, что и аминокислотные остатки CL-домена, сцепленного с Fv-фрагментом легкой цепи домена, связывающего комплекс Т-клеточного рецептора, и аминокислотные остатки СН1-домена, сцепленного с Fv-фрагментом тяжелой цепи антигенсвязывающего домена, имеют электрические заряды, противоположные зарядам аминокислотных остатков CL-домена, сцепленного с Fv-фрагментом легкой цепи антигенсвязывающего домена.
Для создания альтернативного варианта структуры с контролируемой ассоциацией можно получать полипептидный комплекс, в котором аминокислотные остатки CH1-домена, сцепленного с Fv-фрагментом тяжелой цепи домена, связывающего комплекс Т-клеточного рецептора, имеют такие же электрические заряды, что и аминокислотные остатки CL-домена, сцепленного с Fv-фрагментом легкой цепи антигенсвязывающего комплекса; аминокислотные остатки CH1-домена, сцепленного с Fv-фрагментом тяжелой цепи домена, связывающего комплекс Т-клеточного рецептора, имеют такие же электрические заряды, что и аминокислотные остатки CL-домена, сцепленного с Fv-фрагментом легкой цепи домена, связывающего комплекс Т-клеточного рецептора; и аминокислотные остатки СРП-домена, сцепленного с Fv-фрагментом тяжелой цепи антигенсвязывающего домена, имеют электрические заряды, противоположные зарядам аминокислотных остатков CL-домена, сцепленного с Fv-фрагментом легкой цепи антигенсвязывающего домена.
Для создания еще одного варианта структуры с контролируемой ассоциацией можно получать полипептидный комплекс, в котором аминокислотные остатки CH1-домена, сцепленного с Fv-фрагментом тяжелой цепи домена, связывающего комплекс Т-клеточного рецептора, имеют такие же электрические заряды, что и аминокислотные остатки CL-домена, сцепленного с Fv-фрагментом легкой цепи антигенсвязывающего домена; аминокислотные остатки СН1-домена, сцепленного с Fv-фрагментом тяжелой цепи антигенсвязывающего домена, имеют такие же электрические заряды, что и аминокислотные остатки CL-домена, сцепленного с Fv-фрагментом легкой цепи домена, связывающего комплекс Т-клеточного рецептора; аминокислотные остатки CH1-домена, сцепленного с Fv-фрагментом тяжелой цепи домена, связывающего комплекс Т-клеточного рецептора, имеют электрические заряды, противоположные зарядам аминокислотных остатков CL-домена, сцепленного с Fv-фрагментом легкой цепи домена, связывающего Т-клеточный рецептор; и аминокислотные остатки CH1-домена, сцепленного с Fv-фрагментом тяжелой цепи антигенсвязывающего домена, имеют электрические заряды, противоположные зарядам аминокислотных остатков CL-домена, сцепленного с Fv-фрагментом легкой цепи антигенсвязывающего домена.
Контроль электрических зарядов СН1- и CL-доменов
Для получения биспецифического полипептидного комплекса, который распознает эпитоп домена, связывающего Т-клеточный рецептор, посредством тяжелых и легких цепей домена, связывающего Т-клеточный рецептор, и эпитоп антигена посредством тяжелых и легких цепей антигенсвязывающего домена, теоретически можно получать десять типов молекул полипептидных комплексов, если при создании полипептидного комплекса экспрессировать каждую из четырех цепей.
Однако требуемую молекулу полипептидного комплекса предпочтительно можно получать, например, осуществляя такой контроль доменов, который приводит к ингибированию ассоциации между тяжелой цепью домена, связывающего Т-клеточный рецептор, и легкой цепью антигенсвязывающего домена и/или ассоциацию между тяжелой цепью антигенсвязывающего домена и легкой цепью домена, связывающего Т-клеточный рецептор.
Примерами являются изменения аминокислотных остатков, которые образуют поверхность раздела между СН1 тяжелой цепи домена, связывающего Т-клеточный рецептор, и CL легкой цепи антигенсвязывающего домена, на положительно заряженные аминокислотные остатки, и изменения аминокислотных остатков, которые образуют поверхность раздела между СН1 тяжелой цепи и CL легкой цепи домена, связывающего Т-клеточный рецептор, на отрицательно заряженные аминокислотные остатки. В результате таких изменений нежелательная ассоциация между СН1 тяжелой цепи домена, связывающего Т-клеточный рецептор, и CL легкой цепи антигенсвязывающего домена ингибируется, поскольку электрические заряды аминокислотных остатков, которые образуют поверхность раздела, в обоих случаях являются положительными, и нежелательная ассоциация между СН1 тяжелой цепи антигенсвязывающего домена и CL легкой цепи домена, связывающего Т-клеточный рецептор, также ингибируется, поскольку электрические заряды аминокислотных остатков, которые образуют поверхность раздела, в обоих случаях являются отрицательными. Требуемый полипептидный комплекс, предлагаемый в настоящем изобретении, можно эффективно создавать в результате требуемой ассоциации между СН1 тяжелой цепи домена, связывающего Т-клеточный рецептор, и CL легкой цепи домена, связывающего Т-клеточный рецептор, а также в результате требуемой ассоциации между СН1 тяжелой цепи антигенсвязывающего домена и CL легкой цепи антигенсвязывающего домена. Кроме того, требуемая ассоциация между тяжелыми и легкими цепями домена, связывающего Т-клеточный рецептор, предпочтительно усиливается, поскольку аминокислотные остатки, которые образуют поверхность раздела, имеют противоположные друг другу электрические заряды. Требуемая ассоциация между тяжелыми и легкими цепями антигенсвязывающего домена также предпочтительно усиливается, поскольку аминокислотные остатки, которые образуют поверхность раздела, имеют противоположные друг другу электрические заряды. Это позволяет эффективно создавать полипептидный комплекс, предлагаемый в настоящем изобретении, с требуемой ассоциацией.
Кроме того, контроль ассоциации, предлагаемой в настоящем изобретении, можно использовать также для ингибирования ассоциации между СН1-доменами (тяжелых цепей домена, связывающего Т-клеточный рецептор, и антигенсвязывающего домена) или между CL-доменами (легких цепей домена, связывающего Т-клеточный рецептор, и антигенсвязывающего домена).
Специалисты в данной области могут соответствующим образом отбирать требуемый полипептидный комплекс, в котором ассоциация контролируется согласно настоящему изобретению типом аминокислотных остатков, локализованных в непосредственной близости к поверхности раздела CH1/CL при ассоциации.
Кроме того, с использованием публичных баз данных или т.п.специалисты в данной области могут соответствующим образом отбирать приемлемые последовательности СН1 и CL антител в организме, таком как человек, обезьяна, мышь или кролик. Более конкретно, информацию об аминокислотных последовательностях СН1 и CL можно получить с помощью методов, описанных ниже в примерах.
В частности, как продемонстрировано в описанных ниже примерах, конкретные примеры комбинаций аминокислотных остатков, локализованных в непосредственной близости (граничащих или контактирующих) на поверхности раздела CH1/CL при ассоциации между СН1 и CL, которые соответствующим образом сцеплены с VH и VL, образующими домен, связывающий Т-клеточный рецептор, или антигенсвязывающий домен, включают:
- лизин (K) в положении 147 (EU-нумерация) в СН1 (например, в положении 147 в аминокислотной последовательности SEQ ID NO: 1) и треонин (Т) в положении 180 (EU-нумерация) в граничащем (контактирующем) CL;
- лизин (K) в положении 147 (EU-нумерация) в СН1 и серии (S) в положении 131 (EU- нумерация) в граничащем (контактирующем) CL;
- лизин (K) в положении 147 (EU-нумерация) в СН1 и треонин (Т) в положении 164 (EU-нумерация) в граничащем (контактирующем) CL;
- лизин (K) в положении 147 (EU-нумерация) в СН1 и аспарагин (N) в положении 138 (EU-нумерация) в CL, которые граничат (контактируют) друг с другом;
- лизин (K) в положении 147 (EU-нумерация) в СН1 и глутаминовая кислота (Е) в положении 123 (EU-нумерация) в граничащем (контактирующем) CL;
- глутамин (Q) в положении 175 (EU нумерация) в СН1 и глутамин (Q) в положении 160 (EU-нумерация) в граничащем (контактирующем) CL;
или
- лизин (K) в положении 213 (EU-нумерация) в СН1 и глутаминовая кислота (Е) в положении 123 (EU-нумерация) в граничащем (контактирующем) CL.
Эти положения пронумерованы согласно руководству Кэбота с соавторами. (Kabat Е.А. и др., Sequence of Proteins of Immunological Interest. NIH, 1991).
В контексте настоящего описания номера, обозначенные с помощью EU-индекса, соответствуют EU-нумерации (Sequences of proteins of immunological interest, NIH Publication №91-3242). Согласно настоящему изобретению понятия «аминокислотный остаток в положении X (EU-нумерация)» и «аминокислота в положении X (EU-нумерация)» (где X обозначает произвольный номер) применяют взаимозаменяемо с понятиями «аминокислотный остаток, соответствующий положению X (EU-нумерация)», «аминокислота, соответствующая положению X (EU-нумерация)».
Как описано ниже в примерах, требуемый полипептидный комплекс предпочтительно можно получать, осуществляя замену аминокислотных остатков и применяя способы, предлагаемые в настоящем изобретении.
В отношении указанных выше аминокислотных остатков известно, что они являются высококонсервативными у человека и мыши (J. Mol. Recognit. 16, 2003, сс.113-120). Таким образом, изменяя аминокислотные остатки, соответствующие указанным выше аминокислотным остаткам, можно контролировать также ассоциацию между СН1 и CL в константной области полипептидного комплекса, предлагаемого в настоящем изобретении, который отличается от полипептидных комплексов, описанных в примерах.
В частности, в настоящем изобретении предложены полипептидные комплексы с контролируемой ассоциацией между тяжелой цепью и легкой цепью, в которых одна, две или большее количество пар, выбранных из группы, состоящей из пар аминокислотных остатков, указанных ниже в подпунктах (а)-(е), имеют одинаковые электрические заряды:
(а) аминокислотный остаток в положении 147 (EU-нумерация) в СН1 и аминокислотный остаток в положении 180 (EU нумерация) в CL;
(б) аминокислотный остаток в положении 147 (EU-нумерация) в СН1 и аминокислотный остаток в положении 131 (EU-нумерация) в CL;
(в) аминокислотный остаток в положении 147 (EU-нумерация) в СН1 и аминокислотный остаток в положении 164 (EU-нумерация) в CL;
(г) аминокислотный остаток в положении 147 (EU-нумерация) в СН1 и аминокислотный остаток в положении 138 (EU-нумерация) в CL;
(д) аминокислотный остаток в положении 147 (EU-нумерация) в СН1 и аминокислотный остаток в положении 123 (EU-нумерация) в CL и
(е) аминокислотный остаток в положении 175 (EU-нумерация) в СН1 и аминокислотный остаток в положении 160 (EU-нумерация) в CL.
Кроме того, другим вариантом осуществления настоящего изобретения являются антитела, в которых аминокислотные остатки в паре аминокислотных остатков, указанных ниже в подпункте (ж), имеют одинаковые электрические заряды:
(ж) аминокислотный остаток в положении 213 (EU-нумерация) в СН1 и аминокислотный остаток в положении 123 (EU-нумерация) в CL.
Аминокислотные остатки в каждой из указанных выше пар локализуются при ассоциации в непосредственной близости друг к другу, что описано ниже в примерах. С помощью методов моделирования на основе гомологии или других методов с использованием поступающих в продажу программ, специалисты в данной области могут соответствующим образом выявлять в требуемых СН1 или CL положения аминокислот, соответствующие аминокислотным остаткам, указанным выше в подпунктах (а)-(ж), и могут соответствующим образом изменять аминокислотные остатки в этих положениях.
Указанный «несущий электрический заряд аминокислотный остаток» в описанном выше антителе предпочтительно выбирают, например, из аминокислотных остатков, принадлежащих к указанной ниже группе (X) или (Y):
(X) глутаминовая кислота (Е) и аспарагиновая кислота (D); и (Y) лизин (К), аргинин (R), гистидин (Н).
Касательно описанных выше полипептидных комплексов понятие «имеющий такой же электрический заряд» означает, например, что все из двух или большего количества аминокислотных остатков принадлежат к одной из описанных выше групп (X) и (Y). С другой стороны, понятие «имеет противоположный электрический заряд» означает, например, что по меньшей мере один из двух или большего количества аминокислотных остатков представляет собой аминокислотный остаток, принадлежащий к одной из описанных выше групп (X) и (Y), а другие аминокислотные остатки представляют собой аминокислотные остатки, которые принадлежат к другой группе.
Предпочтительными вариантами осуществления настоящего изобретения являются также способы получения вышеописанных полипептидных комплексов и способы, предлагаемые в настоящем изобретении, контроля ассоциации путем замены аминокислотных остатков из групп, указанных выше в подпунктах (а)-(ж), на аминокислотные остатки, которые имеют такой же электрический заряд.
В контексте настоящего изобретения «подлежащие замене» аминокислотные остатки не ограничены указанными выше аминокислотными остатками константной области. С помощью метода моделирования на основе гомологии или других методов, основанных на использовании поступающих в продажу программ, специалисты в данной области могут соответствующим образом идентифицировать аминокислотные остатки, которые образуют поверхность раздела в мутантном полипептиде или гетеромерном мультимере, и соответствующим образом заменять аминокислотные остатки в таких положениях с целью контроля ассоциации.
В методах ингибирования нежелательной ассоциации между тяжелой цепью и легкой цепью путем интродукции вызывающих отталкивание зарядов на поверхности раздела между вариабельными областями тяжелой цепи и легкой цепи, аминокислотные остатки, контактирующие друг с другом на поверхности раздела между вариабельной областью тяжелой цепи (VH) и вариабельной областью легкой цепи (VL), представляют собой, например, глутамин (Q) в положении 39 (например, в положении 39 в аминокислотной последовательности SEQ ID NO: 6, описанной в WO 2006/106905) в вариабельной области тяжелой цепи FR2 и глутамин (Q) в положении 38 (например, в положении 44 в аминокислотной последовательности SEQ ID NO: 8, описанной в WO 2006/106905) в граничащей (контактирующей) вариабельной области легкой цепи FR2. Такие предпочтительные аминокислотные остатки представляют собой также, например, лейцин (L) в положении 45 (например, в положении 45 в аминокислотной последовательности SEQ ID NO: 6, описанной в WO 2006/106905) в вариабельной области тяжелой цепи FR2 и пролин (Р) в положении 44 (например, в положении 44 в аминокислотной последовательности SEQ ID NO: 8, представленной в WO 2006/106905) в граничащей (контактирующей) вариабельной области легкой цепи FR2. Указанные положения пронумерованы согласно руководству Кэбота соавторами (Kabat Е.А. и др., Sequence of Proteins of Immunological Interest, изд-во NIH, 1991).
В отношении указанных выше аминокислотных остатков известно, что они являются высококонсервативными у человека и мыши (J. Mol. Recognit. 16, 2003, cc. 113-120). Таким образом, изменяя аминокислотные остатки, соответствующие указанным выше аминокислотным остаткам, можно контролировать также ассоциацию между вариабельными областями VH и VL антител, отличающихся от полипептидных комплексов, описанных в примерах.
Более конкретно, указанные антитела, которые имеют вариабельные области тяжелых цепей и легких цепей, включают антитела, в которых указанные ниже аминокислотные остатки (1) и (2) или (3) и (4) имеют одинаковые электрические заряды:
(1) аминокислотный остаток, соответствующий положению 39 (EU-нумерация) в вариабельной области тяжелой цепи;
(2) аминокислотный остаток, соответствующий положению 38 (EU-нумерация) в вариабельной области легкой цепи;
(3) аминокислотный остаток, соответствующий положению 45 (EU-нумерация) в вариабельной области тяжелой цепи;
(4) аминокислотный остаток, соответствующий положению 44 (EU-нумерация) в вариабельной области легкой цепи.
Указанные выше аминокислотные остатки (1) и (2) или (3) и (4) локализуются при ассоциации в непосредственной близости друг к другу. С помощью метода моделирования на основе гомологии или других методов с использованием поступающих в продажу программ, специалисты в данной области могут соответствующим образом идентифицировать в требуемой вариабельной области тяжелой цепи или легкой цепи аминокислотные положения, соответствующие аминокислотным остаткам, указанным выше в ((4), и может соответствующим образом изменять аминокислотные остатки в указанных положениях.
В указанном выше антителе «несущий электрический заряд аминокислотный остаток» предпочтительно выбирают, например, из аминокислотных остатков, принадлежащих к указанной ниже группе (X) или (Y):
(X) глутаминовая кислота (Е) и аспарагиновая кислота (D); и (Y) лизин (K), аргинин (R), гистидин (Н).
В человеческих и мышиных антителах, как правило, указанные выше аминокислотные остатки (1)-(4) представляют собой:
(1) глутамин (Q);
(2) глутамин (Q);
(3) лейцин (L) и
(4) пролин (Р) соответственно.
В предпочтительных вариантах осуществления настоящего изобретения указанные выше аминокислотные остатки изменяют (например, заменяют на имеющую заряд аминокислоту). Типы перечисленных выше аминокислотных остатков (1)-(4) не ограничены указанными выше. Эти аминокислоты могут представлять собой любые другие аминокислоты, соответствующие указанным выше. Например, касательно человеческого антитела, аминокислота, соответствующая аминокислоте в положении 38 (EU-нумерация) в вариабельной области легкой цепи, может представлять собой гистидин (Н). На основе опубликованных документов (например, J. Mol. Recognit. 16, 2003, сс.113-120) или т.п.специалисты в данной области могут определять тип аминокислотного остатка, который соответствует любому положению в легкой цепи, и следовательно могут соответственно изменять аминокислотный остаток (например, осуществлять замену на имеющую заряд аминокислоту).
В методиках ингибирования нежелательной ассоциации между тяжелой цепью и легкой цепью путем замены аминокислотных остатков, которые образуют гидрофобное ядро на поверхности раздела между вариабельными областями тяжелой цепи и легкой цепи, на несущие электрический заряд полярные аминокислоты предпочтительные аминокислотные остатки, обладающие способностью образовывать гидрофобное ядро на поверхности раздела между вариабельной областью тяжелой цепи (VH) и вариабельной области легкой цепи (VL), представляют собой лейцин (L) в положении 45 в вариабельной области тяжелой цепи и пролин (Р) в положении 44, граничащий в вариабельной областью легкой цепи.
В целом, понятие «гидрофобное ядро» относится к участку, в котором боковые цепи гидрофобных аминокислот объединены внутри ассоциированного полипептида. К гидрофобным аминокислотам относятся, например, аланин, изолейцин, лейцин, метионин, фенилаланин, пролин, триптофан и валин. Однако в образовании гидрофобного ядра могут принимать участие аминокислотные остатки, отличные от указанных гидрофобных аминокислот (например, тирозин). В сочетании с гидрофильной поверхностью, образованной выступающими боковыми цепями гидрофильных аминокислот, гидрофобное ядро служит в качестве «движущей силы», ускоряющей ассоциацию водорастворимых полипептидов. Когда гидрофобные аминокислоты двух различных доменов присутствуют на поверхности молекулы и являются доступными для молекул воды, то энтропия возрастает, что приводит к увеличению свободной энергии. В результате ассоциация двух доменов друг с другом должна быть такой, чтобы снижать свободную энергию, что требуется для стабилизации. Гидрофобные аминокислоты по поверхности раздела погружаются внутрь молекулы с образованием гидрофобного ядра.
Считается, что, когда гидрофобные аминокислоты, которые образуют гидрофобное ядро при ассоциации полипептидов, изменяют на полярные аминокислоты, несущие электрический заряд, то образование гидрофобного ядра ингибируется. Это приводит к ингибированию ассоциации полипептидов.
Для создания полипептидных комплексов, предлагаемых в настоящем изобретении, можно применять также другие известные методики. Например, для усиления ассоциации первой VH (VH1) и первой VL (VL1) и/или второй VH (VH2) и второй VL (VL2), помимо «изменений», предлагаемых в настоящем изобретении, аминокислоты в одной из вариабельных областей Н-цепи заменяют на аминокислоты, которые имеют более крупную боковую цепь («выступ»; «выпуклость»), а аминокислоты в вариабельной области другой Н-цепи заменяют на аминокислоты с боковой цепью меньшего размера («впадина»; «пустота»), в результате чего «выступ» помещается во «впадину». Это усиливает ассоциацию VH1 и VL1 и/или VH2 и VL2, что приводит к дополнительному ингибированию ассоциации между полипептидами VH1 и VL2 и/или между VH2 и VL1 (WO 1996/027011; Ridgway J.B. и др., Protein Engineering 9, 1996, cc. 617-621; Merchant A.M. и др., Nature Biotechnology 16, 1998, cc. 677-681).
При создании описанного выше полипептидного комплекса каждый домен можно соединять непосредственно через пептидную связь или осуществлять связывание пептидов через пептидный линкер. В этом случае применяемый линкер может представлять собой линкер, описанный выше в качестве примера, и соответствующие линкеры с пептидной меткой, например, с His-меткой, НА-меткой, myc-меткой или FLAG-меткой. Кроме того, предпочтительно можно использовать способность к взаимному связыванию на основе водородных связей, дисульфидной связи, ковалентной связи или на основе ионного взаимодействия, или их комбинации. Например, можно использовать аффинность между СН1 и CL антитела, или описанные выше Fc-домены, выведенные из биспецифического антитела, можно применять для гетеромерной ассоциации Fc-доменов. Кроме того, предпочтительно можно использовать междоменные дисульфидные мостики, как это описано в примерах.
Полипептидные комплексы, предлагаемые в настоящем изобретении, включают, например, варианты, которые представлены на фиг.17, 19 и 24.
Полипептидные комплексы, предлагаемые в настоящем изобретении, можно получать теми же самыми методами, что и методы, описанные выше для получения рекомбинантных антител.
Кроме того, настоящее изобретение относится к полинуклеотидам, кодирующим полипептидный комплекс, предлагаемый в настоящем изобретении. Полипептидный комплекс, предлагаемый в настоящем изобретении, можно встраивать в любые экспрессионные векторы. Для получения клеток, экспрессирующих полипептидный комплекс, соответствующего хозяина трансформируют экспрессионным вектором. Полипептидный комплекс, кодируемый полинуклеотидом, можно получать путем культивирования клеток, экспрессирующих полипептидный комплекс, и сбора продукта экспрессии из супернатанта культуры. В частности, настоящее изобретение относится к векторам, несущим полинуклеотид, который кодирует полипептидный комплекс, предлагаемый в настоящем изобретении, клеткам, которые содержат векторы, и способам получения полипептидного комплекса, предлагаемого в настоящем изобретении, заключающимся в том, что культивируют клетки и собирают полипептидный комплекс из супернатанта культуры. Для описанных выше целей можно использовать такие же методики, которые описаны выше для рекомбинантных антител.
Фармацевтическая композиция
Другим объектом настоящего изобретения являются фармацевтические композиции, которые содержат в качестве действующего вещества полипептидный комплекс, который включает:
(1) антигенсвязывающий домен;
(2) домен, содержащий Fc-домен с пониженной активностью связывания с Fcγ-рецептором и
(3) CD3-связывающий домен.
Настоящее изобретение относится также к терапевтическим агентам для индукции клеточной цитотоксичности, которые содержат в качестве действующего вещества описанный выше комплекс (терапевтические агенты для индукции клеточной цитотоксичности), агенты, подавляющие рост клеток, и противораковые агенты. Фармацевтические композиции, предлагаемые в настоящем изобретении, можно применять в качестве средства для осуществления терапии или профилактики рака. Согласно настоящему изобретению терапевтические агенты для индукции клеточной цитотоксичности, агенты, подавляющие рост клеток, и противораковые агенты, предлагаемые в настоящем изобретении, предпочтительно вводят индивидуумам, страдающим раком или имеющим вероятность рецидива рака.
Согласно настоящему изобретению терапевтические агенты для индукции клеточной цитотоксичности, агенты, подавляющие рост клеток, и противораковые агенты, которые содержат в качестве действующего вещества полипептидный комплекс, который включает:
(1) антигенсвязывающий домен;
(2) домен, содержащий Fc-домен с пониженной активностью связывания с Fcγ-рецептором и
(3) CD3-связывающий домен,
можно рассматривать также в качестве способа предупреждения или лечения рака, который заключается в том, что осуществляют стадию, на которой вводят полипептидный комплекс индивидууму, или полипептидный комплекс можно применять для приготовления терапевтических агентов для индукции клеточной цитотоксичности, агентов, подавляющих рост клеток, или противораковых агентов.
В контексте настоящего описания понятие «содержат в качестве действующего вещества полипептидный комплекс, который включает:
(1) антигенсвязывающий домен;
(2) домен, содержащий Fc-домен с пониженной активностью связывания с Fcγ-рецептором и
(3) CD3-связывающий домен»,
означает, что композиция содержит полипептидный комплекс в качестве основного действующего вещества; однако относительное содержание полипептидного комплекса конкретно не указано.
Фармацевтическую композицию, предлагаемую в настоящем изобретении, терапевтический агент для индукции клеточной цитотоксичности, агент, подавляющий рост клеток, или противораковый агент, предлагаемый в настоящем изобретении, можно приготавливать при необходимости с использованием различных типов полипептидных комплексов. Например, цитотоксическое действие в отношении клеток, экспрессирующих антиген, можно усиливать с использованием смеси из множества полипептидных комплексов, предлагаемых в настоящем изобретении, которые связываются с одним и тем же антигеном. Альтернативно этому, терапевтическое действие можно повышать путем приготовления полипептидного комплекса, предлагаемого в настоящем изобретении, содержащего антигенсвязывающий домен, который связывается с раковым антигеном, в комбинации с другими полипептидными комплексами, предлагаемыми в настоящем изобретении, который содержат антигенсвязывающий домен для другого антигена.
При необходимости полипептидные комплексы, предлагаемые в настоящем изобретении, можно капсулировать с микрокапсулы (микрокапсулы, изготовленные из гидроксиметилцеллюлозы, желатина, поли(метилметакрилата) и т.п.), и можно включать в компоненты коллоидных систем, предназначенных для введения лекарственных веществ (липосомы, альбуминовые микросферы, микроэмульсии, наночастицы и нанокапсулы) (например, см. «Remington's Pharmaceutical Science 16-ое изд.», под ред. Oslo, 1980)). Кроме того, известны методы приготовления агентов в виде средств с замедленным высвобождением, и их можно применять для полипептидных комплексов, предлагаемых в настоящем изобретении (J. Biomed. Mater. Res. 15, 1981, сс.267-277; Chemtech. 12, 1982, сс.98-105; US №3773719; опубликованные европейские патенты (ЕР) ЕР 58481 и ЕР 133988; Biopolymers 22, 1983, сс.547-556).
Фармацевтические композиции, агенты, подавляющие рост клеток, или противораковые агенты, предлагаемые в настоящем изобретении, можно вводить пациенту либо орально, либо парентерально. Предпочтительным является парентеральное введение. В частности, указанные методы введения включают инъекцию, введение в нос, транспульмонарное введение и чрескожное введение. Инъекции включают, например, внутривенные инъекции, внутримышечные инъекции, внутрибрюшинные инъекции и подкожные инъекции. Например, фармацевтические композиции, терапевтические агенты для индукции клеточной цитотоксичности, агенты, подавляющие рост клеток, или противораковые агенты, предлагаемые в настоящем изобретении, можно применять местно или системно путем инъекции. Кроме того, приемлемые методы введения можно выбирать в зависимости от возраста пациента и симптомов. Вводимую дозу можно выбирать, например, из следующего диапазона: от 0,0001 до 1000 мг на кг веса тела на каждое введение. Альтернативно этому, дозу можно выбирать, например, из следующего диапазона: от 0,001 до 100000 мг/пациента. Однако доза фармацевтической композиции, предлагаемой в настоящем изобретении, не ограничена указанными дозами.
Фармацевтические композиции, предлагаемые в настоящем изобретении, можно приготавливать с помощью общепринятых методов (например, см. Remington's Pharmaceutical Science, последнее изд., изд-во Mark Publishing Company, Easton, U.S.A.), и они могут содержать также фармацевтически приемлемые носители и добавки. Их примерами являются (но, не ограничиваясь только ими) поверхностно-активные вещества, эксципиенты, красители, ароматизаторы, консерванты, стабилизаторы, буферы, суспендирующие агенты, придающие изотоничность агенты, связующие вещества, разрыхлители, замасливатели, повышающие текучесть агенты и корригенты и можно применять другие общепринятые носители. Конкретными примерами носителей являются легкая безводная кремниевая кислота, лактоза, кристаллическая целлюлоза, маннит, крахмал, кармеллоза кальция, кармеллоза натрия, гидроксипропилцеллюлоза, гидроксипропилметилцеллюлоза, поливинилацетальдиэтиламиноацетат, поливинилпирролидон, желатин, триглицерид со средней длиной цепи, полиоксиэтиленированное гидрогенизированное касторовое масло 60, сахароза, карбоксиметилцеллюлоза, кукурузный крахмал, неорганическая соль и т.п.
В настоящем изобретении предложены также способы повреждения клеток, экспрессирующих раковый антиген, или подавления клеточного роста, заключающиеся в том, что приводят в контакт клетки, экспрессирующие раковый антиген, с полипептидным комплексом, предлагаемым в настоящем изобретении, который связывается с раковым антигеном. Моноклональные антитела, которые связываются с раковым антигеном, описаны выше как связывающий раковый антиген полипептидный комплекс, предлагаемый в настоящем изобретении, который входит в состав терапевтических агентов для индукции клеточной цитотоксичности, агентов, подавляющих рост клеток, или противораковых агентов, предлагаемых в настоящем изобретении. Изобретение не ограничено клетками, с которыми связывается связывающий раковый антиген полипептидный комплекс, предлагаемый в настоящем изобретении, если они экспрессируют раковый антиген. В частности, в настоящем изобретении предпочтительными экспрессирующими раковый антиген клетками являются клетки рака яичника, клетки рака предстательной железы, клетки рака молочной железы, клетки рака матки, клетки рака печени, клетки рака легкого, клетки рака поджелудочной железы, клетки рака желудка, клетки рака мочевого пузыря и клетки рака ободочной кишки. Если раковый антиген представляет собой GPC3, то изобретение не ограничено конкретными клетками, если они экспрессируют GPC3. Однако предпочтительными раковыми клетками являются клетки гепатокарциномы, клетки рака легкого и клетки рака яичника.
Согласно настоящему изобретению «приведение в контакт» можно осуществлять, например, добавляя связывающий раковый антиген полипептидный комплекс, предлагаемый в изобретении, к культуральным средам клеток, экспрессирующих раковый антиген, которые культивируют in vitro. В этом случае добавляемый полипептидный комплекс можно применять в пригодной для этой цели форме, такой как раствор или твердое вещество, полученное путем лиофилизации, или т.п. Когда полипептидный комплекс, предлагаемый в настоящем изобретении, добавляют в виде водного раствора, то раствор может представлять собой чистый водный раствор, содержащий только полипептидный комплекс, или раствор, содержащий, например, указанные выше поверхностно-активное вещество, эксципиент, краситель, ароматизатор, консервант, стабилизатор, буфер, суспендирующий агент, придающий изотоничность агент, связующее вещество, разрыхлитель, замасливатель, повышающий текучесть агент и корргиент.Концентрация добавляемого комплекса не имеет решающего значения; однако конечная концентрация в культуральной среде предпочтительно составляет от 1 пг/мл до 1 г/мл, более предпочтительно от 1 нг/мл до 1 мг/мл и еще более предпочтительно от 1 мкг/мл до 1 мг/мл.
Согласно другому варианту осуществления настоящего изобретения «приведение в контакт» можно осуществлять также in vivo путем обработки животных кроме человека, которым трансплантированы экспрессирующие раковый антиген клетки, или животных, у которых эндогенно происходит экспрессия ракового антигена. Метод введения может быть оральным или парентеральным. Предпочтительным является парентеральное введение. В частности, указанные методы введения включают инъекцию, введение в нос, введение в легкое и чрескожное введение. Инъекции включают, например, внутривенные инъекции, внутримышечные инъекции, внутрибрюшинные инъекции и подкожные инъекции. Например, фармацевтические композиции, терапевтические агенты для индукции клеточной цитотоксичности, агенты, подавляющие рост клеток, или противораковые агенты, предлагаемые в настоящем изобретении, можно применять местно или системно путем инъекции. Кроме того, приемлемые методы введения можно выбирать в зависимости от возраста животного и симптомов. Когда полипептидный комплекс, предлагаемый в настоящем изобретении, добавляют в виде водного раствора, то раствор может представлять собой чистый водный раствор, содержащий только полипептидный комплекс, или раствор, содержащий, например, указанные выше поверхностно-активное вещество, эксципиент, краситель, ароматизатор, консервант, стабилизатор, буфер, суспендирующий агент, придающий изотоничность агент, связующее вещество, разрыхлитель, замасливатель, повышающий текучесть агент и корригент.Вводимую дозу можно выбирать, например, из следующего диапазона: от 0,0001 до 1000 мг на кг веса тела на каждое введение. Альтернативно этому, дозу можно выбирать, например, из следующего диапазона: от 0,001 до 100000 мг/пациента. Однако доза полипептидного комплекса, предлагаемого в настоящем изобретении, не ограничена указанными примерами.
Описанные ниже методы предпочтительно используют в качестве метода оценки или определения клеточной цитотоксичности, обусловленной контактированием полипептидного комплекса, предлагаемого в настоящем изобретении, с экспрессирующими антиген клетками, с которыми связывается антигенсвязывающий домен, входящий в полипептидный комплекс, предлагаемый в настоящем изобретении. Методы оценки или определения цитотоксической активности in vitro включают методы определения активности цитотоксических Т-клеток или т.п. Обладает ли полипептидный комплекс, предлагаемый в настоящем изобретении, способностью индуцировать опосредуемую Т-клетками клеточную цитотоксичность, можно определять с помощью известных методов (см., например, Current protocols in Immunology, глава 7. Immunologic studies in humans, под ред. John E, Coligan и др., изд-во, John Wiley & Sons, Inc., 1993). При осуществлении анализа цитотоксичности полипептидный комплекс, антигенсвязывающий домен которого связывается с антигеном, отличным от антигена, распознаваемого антигенсвязывающим доменом полипептидного комплекса, предлагаемого в настоящем изобретении, и который не экспрессируется на клетках, применяют в качестве контрольного полипептидного комплекса. Контрольный полипептидный комплекс анализируют таким же образом. Затем оценивают активность, определяя, обладает ли полипептидный комплекс, предлагаемый в настоящем изобретении, более выраженной цитотоксической активностью, чем контрольный полипептидный комплекс.
Цитотоксическую активность in vivo оценивают или определяют, например, с помощью следующей процедуры. Клетки, экспрессирующие антиген, с которым связывается антигенсвязывающий домен, входящий в полипептидный комплекс, предлагаемый в настоящем изобретении, трансплантируют внутрикожно или подкожно животному кроме человека. Затем, начиная со дня трансплантации или после него, вводят тестируемый полипептидный комплекс в вену или перитонеальную полость каждый день или с интервалами в несколько дней. В течение времени определяют размер опухоли. Различие в изменении размера опухоли можно рассматривать как цитотоксическую активность. Также как и в анализе in vitro, вводят контрольный полипептидный комплекс.Можно считать, что полипептидный комплекс, предлагаемый в настоящем изобретении, обладает цитотоксической активностью, когда размер опухолей является меньшим в группе, которую обрабатывали полипептидным комплексом, предлагаемым в настоящем изобретении, чем в группе, которую обрабатывали контрольным полипептидным комплексом.
МТТ-метод и измерение меченного с помощью изотопа поглощения тимидина в клетки предпочтительно применяют для оценки или определения воздействия контакта с полипептидным комплексом, предлагаемым в настоящем изобретении, на подавление роста клеток, экспрессирующих антиген, с которым связывается антигенсвязывающий домен, входящий в полипептидный комплекс. При этом методы, аналогичные описанным выше для оценки или определения цитотоксической активности in vivo, предпочтительно можно использовать для оценки или определения способности подавлять рост клеток in vivo.
В настоящем изобретении предложены также наборы, которые можно применять в способе, предлагаемом в настоящем изобретении, которые содержат полипептидный комплекс, предлагаемый в настоящем изобретении, или полипептидный комплекс, полученный способом, предлагаемом в настоящем изобретении. Наборы могут быть упакованы вместе с дополнительным фармацевтически приемлемым носителем или средой, или с инструкцией, включающей руководство по применению наборов и т.д.
Кроме того, настоящее изобретение относится к полипептидным комплексам, предлагаемым в настоящем изобретении, или полипептидным комплексам, полученным способом, предлагаемом в настоящем изобретении, для применения в способе, предлагаемом в настоящем изобретении.
Все процитированные в настоящем описании документы, характеризующие известный уровень техники, включены в настоящее описание в качестве ссылки.
Примеры
Ниже настоящее изобретение описано со ссылкой на конкретные примеры, которые не ограничивают объем изобретения.
Пример 1. Конструирование и изучение GPC3 ERY2
(1) Основные принципы
Существует хорошо известный метод пролонгирования времени полужизни в крови вводимого белка in vivo, который основан на опосредуемой FcRn рециркуляции с использованием представляющего интерес белка, конъюгированного с Fc-доменом антитела. Однако конъюгация Fc естественного типа с BiTE может приводить к индукции различных цитокинов, поскольку одна и та же молекула может связываться с Т-клеткой через анти-CD3 scFv-фрагмент ее BiTE-остатка и одновременно связываться с FcgR (Fcγ-рецептор) на клеточной мембране, например, NK-клетки или макрофага, через ее Fc-домен, и образовавшаяся перекрестная сшивка может активировать эти клетки независимым от ракового антигена образом. Таким образом, получали молекулу, обозначенную как ERY2, в которой антитело в виде BiTE сцеплено через полипептидный линкер с Fc-доменом, обладающим пониженной активностью связывания с Fcγ- рецептором (молчащий Fc), и активность ERY2 оценивали путем сравнения с активностью BiTE. scFv антитела к эпсилон цепи CD3, соединяли через короткий пептидный линкер с scFv антитела к глипикану 3 (GPC3), который представляет собой заякоренный с помощью GPI белок, для которого известен высокий уровень экспрессии на клетках рака печени, с получением BiTE к GPC3 (GPC3 BiTE) (фиг.17А). Затем его соединяли с молчащим Fc с получением ERY2 к GPC3 (GPC3 ERY2) (фиг.17В). Кроме того, для сравнения создавали антитела к GPC3 обычного IgG-типа. Антитело к GPC3 IgG-типа получали в виде антитела с пониженным содержанием фукозы во фрагменте сахарной цепи, т.е. антитела с низким уровнем фукозы, которое, как известно, обладает повышенной ADCC-активностью.
(2) Конструирование GPC3 BiTE
С помощью ПЦР-амплификации с использованием в качестве матрицы экспрессионного вектора для антитела к GPC3 получали кДНК, каждая из которых кодировала либо вариабельную область Н-цепи (анти-GPC3 VH), либо вариабельную область L-цепи (анти-GPC3 VL). ПЦР осуществляли с помощью праймеров, содержащих соответствующие дополнительные последовательности, и указанных выше кДНК в качестве матриц для конструирования кДНК-фрагмента, кодирующего анти-GPC3 scFv, имеющего аминокислотную последовательность, в которой анти-GPC3 VH и анти-GPC3 VL сцеплены с помощью линкера с тремя повторами Gly-Gly-Gly-Gly-Ser (SEQ ID NO: 7).
Кроме того, получали серии олигонуклеотидов, каждый из которых имел нуклеотидную последовательность, кодирующую частичную последовательность вариабельной области Н-цепи (М12 VH) или вариабельную область L-цепи (М12 VL) антитела к CD3 (M12), и имел на концах комплементарные последовательности. Олигонуклеотиды создавали таким образом, чтобы их можно было связывать друг с другом через области комплементарных последовательностей с помощью полимеразной реакции для синтеза полинуклеотида, соответствующего вариабельной области Н-цепи (M12 VH) и вариабельной области L-цепи (М12 VL). Олигонуклеотиды смешивали и затем связывали друг с другом с помощью ПЦР с получением двух кДНК, кодирующих аминокислотные последовательности соответствующих вариабельных областей. ПЦР осуществляли с помощью праймеров, содержащих соответствующие дополнительные последовательности, и указанных выше кДНК в качестве матриц для конструирования кДНК-фрагмента, кодирующего scFv М12, имеющего аминокислотную последовательность, в которой М12 VL и М12 VH сцеплены через линкер, имеющий три повтора Gly-Gly-Gly-Gly-Ser (SEQ ID NO: 7).
Затем, с помощью ПЦР с использованием праймеров, содержащих соответствующие дополнительные последовательности, и указанных выше кДНК-фрагментов, каждый из которых кодировал либо анти-GPC3 scFv, либо М12 scFv, в качестве матриц, конструировали кДНК-фрагмент, который кодировал аминокислотную последовательность, в которой анти-GPC3 scFv и М12 scFv соединяли через линкер, состоящий из Gly-Gly-Gly-Gly-Ser (SEQ ID NO: 7), и с His-меткой на С-конце (восемь остатков гистидина) (последовательность SEQ ID NO: 33 без 19 аминокислот на аминоконце).
Осуществляли ПЦР с использованием праймеров, содержащих соответствующие дополнительные последовательности, и в качестве матрицы кДНК-фрагмента, кодирующего аминокислотную последовательность SEQ ID NO: 33, лишенную 19 аминоконцевых аминокислот, для получения кДНК-фрагмента, в котором расщепляемую EcoRl последовательность, последовательность Козак, и нуклеотидную последовательность, кодирующую секреторную сигнальную последовательность, присоединяли к 5'-концу указанного выше кДНК-фрагмента, а расщепляемую NotI последовательность присоединяли к 3'-концу. Образовавшийся кДНК-фрагмент расщепляли с помощью EcoRI и NotI и встраивали в экспрессионный вектор для клеток млекопитающих с получением экспрессионного вектора для GPC3 BiTE (SEQ ID NO: 33; зрелая последовательность не содержит 19 аминокислот на аминоконце, которые служат в качестве сигнальной последовательности).
Вектор интродуцировали в клетки СНО DG44 с помощью электропорации. После серийного разведения клетки культивировали в присутствии 1 мг/мл генетицина для выделения устойчивых к лекарственному средству клеточных линий. Супернатант культуры полученных клеточных линий анализировали с помощью вестерн-блоттинга с использованием антитела к His-метке для отбора клеточных линий, экспрессирующих GPC3 BiTE.
Супернатант культуры, полученный в результате крупномасштабного культивирования указанной клеточной линии, вносили в колонку, заполненную SP Сефарозой FF column (фирма GE Healthcare). После отмывки колонки фракцию, содержащую GPC3 BiTE, элюировали с использованием концентрационного градиента NaCl. Фракцию вносили в колонку HisTrap HP (фирма GE Healthcare). После отмывки колонки фракцию, содержащую GPC3 BiTE, элюировали с использованием концентрационного градиента имидазола. Фракцию концентрировали ультрафильтрацией и затем концентрат вносили в колонку, заполненную Супердекс 200 (фирма GE Healthcare). Собирали только мономерную содержащую GPC3 BiTE фракцию с получением очищенного GPC3 BiTE.
(3) Конструирование GPC3 ERY2
Осуществляли ПЦР с использованием праймеров, содержащих такие же соответствующие дополнительные последовательности, что и в описанном выше методе, и с использованием метода, хорошо известного специалистам в данной области, такого метод сайтнаправленного мутагенеза с использованием набора QuikChange Site-Directed Mutagenesis (фирма Stratagene), для получения экспрессионных векторов, в которые встроен полинуклеотид, кодирующий GPC3 ERY2_Hk (SEQ ID NO: 34; зрелая последовательность, которая не содержит 19 аминокислот на аминоконце, служащих в качестве сигнальной последовательности) или GPC3 ERY2 Hh (SEQ ID NO: 35; зрелая последовательность, которая не содержит 19 аминокислот на аминоконце, служащих в качестве сигнальной последовательности).
Эти экспрессионные векторы совместно интродуцировали в клетки линии FreeStyle293-F (фирма Invitrogen) для кратковременной экспрессии GPC3 ERY2. Образовавшийся супернатант культуры вносили в колонку, содержащую Anti FLAG М2 (фирма Sigma). После отмывки колонку элюировали, используя FLAG пептид в концентрации ОД мг/мл (фирма Sigma). Фракцию, содержащую GPC3 ERY2, вносили в колонку HisTrap HP (фирма GE Healthcare). После отмывки колонку элюировали с использованием концентрационного градиента имидазола. Фракцию, содержащую GPC3 ERY2, концентрировали ультрафильтрацией и затем концентрат вносили в колонку, заполненную Супердекс 200 (фирма GE Healthcare). Собирали только мономерную содержащую GPC3 ERY2 фракцию с получением очищенного GPC3 ERY2.
(4) Конструирование антитела к GPC3 с низким уровнем фукозы
Экспрессионный вектор для антитела к GPC3 (обозначено как гуманизированное антитело GC33 в WO 2006/006693) интродуцировали в клетки с «выключенным» геном ГДФ-фукозы линии СНО DXB11 (Cancer Sci. 101(10), 2010, сс.2227-2233) путем электропорации. После серийного разведения клетки культивировали в присутствии 0,5 мг/мл генетицина для отбора устойчивых к лекарственному средству линий, и получали клеточные линии, экспрессирующие антитело GPC3 с низким уровнем фукозы. Из супернатанта культуры, полученного при культивировании этих клеток, получали содержащую антитело фракцию путем общепринятой очистки посредством аффинной хроматографии с использованием колонки Hitrap®, заполненной белком А (фирма Pharmacia). Затем содержащую антитело фракцию очищали с помощью гель-фильтрации с использованием Супердекс 20026/60 (фирма Pharmacia). Из элюата собирали мономерную фракцию с получением антитела к GPC3 с низким уровнем фукозы.
(5) Анализ цитотоксичности с использованием мононуклеарных клеток периферической крови человека
(5-1) Получение суспензии мононуклеарных клеток периферической крови человека (РВМС)
Получали образцы по 50 мл периферической крови здоровых добровольцев (взрослых) с использованием шприцев, предварительно заполненных 100 мкл (1000 ед./мл) раствора гепарина (Novo-Heparin 5000 ед. на инъекцию; фирма Novo Nordisk). Периферическую кровь двукратно разводили ЗФР(-), разделяли на четыре одинаковые аликвоты и добавляли в пробирки для разделения лимфоцитов Leucosep (каталожный №227290; система Greiner bio-one), в которые предварительно инъецировали 15 мл фиколл-пак PLUS и центрифугировали. После центрифугирования пробирок для разделения (2150 об/мин, 10 мин, комнатная температура) собирали слой фракции мононуклеарных клеток. Клетки во фракции мононуклеарных клеток однократно отмывали с использованием модифицированной по методу Дульбекко среды Игла (фирма SIGMA), содержащей 10% FBS (далее в контексте настоящего описания обозначена как 10%FBS/D-MEM), а затем плотность клеток доводили до 4×106 клеток/мл с помощью 10%FBS/D-MEM. Полученную таким образом клеточную суспензию применяли в качестве суспензии человеческих РВМС в последующих экспериментах.
(5-2) Анализ цитотоксической активности
Цитотоксическую активность оценивали на основе степени ингибирования клеточного роста с помощью анализатора клеток в реальном времени xCELLigence (фирма Roche Diagnostics). Применяемые клетки-мишени представляли собой клеточную линию SK-pca13a, созданную на основе клеточной линии SK-HEP-1, в которой усилили экспрессию человеческого GPC3. Клетки SK-pca13a отделяли от чашек и высевали в планшет Е-Plate 96 (фирма Roche Diagnostics) из расчета 1×104 клеток/лунку (100 мкл/лунку). Затем начинали анализ жизнеспособных клеток с помощью анализатора клеток в реальном времени xCELLigence. На следующий день планшет удаляли из анализатора клеток в реальном времени xCELLigence и в планшет добавляли по 50 мкл каждого полученного антитела в различных концентрациях (0,004, 0,04, 0,4 и 4нМ). После осуществления реакции в течение 15 мин при комнатной температуре добавляли 50 мкл суспензии человеческих РВМС (2×105 клеток/лунку), полученной согласно методу, описанному в разделе (5-1). Клетки вновь помещали в анализатор клеток в реальном времени xCELLigence для начала анализа жизнеспособных клеток. Реакцию проводили в атмосфере 5% газообразного диоксида азота при 37°С. Степень ингибирования роста клеток (%) определяли с помощью приведенной ниже формулы с использованием величины клеточного индекс, определенной через 72 ч после добавления человеческих РВМС. Применяемую для расчета величину клеточного индекса стандартизовали таким образом, что величину клеточного индекса, определенную непосредственно перед добавлением антитела, принимали за 1.
Степень ингибирования роста клеток (%) = (А-Б) × 100/(А-1)
А обозначает среднюю величину клеточного индекса в лунке без антитела (только клетка-мишень и человеческие РВМС), а Б обозначает среднюю величину клеточного индекса в каждой лунке. Измерение осуществляли в трех повторностях.
Цитотоксическую активность GPC3 BiTE, GPC3 ERY2 и антитела к GPC3 IgG-типа оценивали с использованием полученных из человеческой крови РВМС (мононуклеарные клетки периферической крови) в качестве эффекторных клеток. У GPC3 BiTE обнаружена очень сильная активность (фиг.1). Эта активность существенно превышала активность антитела к GPC3 с низким уровнем фукозы. Таким образом, GPC3 BiTE может служить в качестве эффективного терапевтического агента, превышающего по своей активности антитело IgG-типа. С другой стороны, активность GPC3 ERY2 была не столь сильной, как у GPC3 BiTE, хотя она тоже превышала активность антитела к GPC3 IgG-типа. Это позволяет предположить, что добавление только Fc в BiTE не приводит к созданию требуемой молекулы.
Пример 2. Конструирование и оценка GPC3 ERY5. GPC3 ERY6 и GPC3 ERY7
Далее с целью улучшения специфической активности домен, связывающий раковый агент (GPC3), превращали в двухвалентный для повышения активности связывания с раковой клеткой. К GPC3 ERY2 добавляли другой анти-GPC3 scFv, создавая GPC3 ERY5 (фиг.17Г). Кроме того, вместо scFv добавляли GPC3-связывающий домен Fab-типа, создавая GPC3 ERY7 (фиг.17Е). Кроме того, конструировали также GPC3 ERY6 (фиг.17Д), в котором scFv к эпсилон-цепи CD3 GPC3 ERY5 расщепляли на два плеча.
Конкретно, для получения серий экспрессионных векторов, в которые встраивали полинуклеотид, кодирующий GPC3 ERY5 Hh, GPC3 ERY6 Hk, GPC3 ERY6 Hh, GPC3 ERY7 Hh или GPC3 ERY7 L, применяли известный специалистам в данной области метод, такой как ПЦР с использованием праймеров, содержащих такие же соответствующие дополнительные последовательности, что и в описанном выше методе.
Для кратковременной экспрессии каждой сконструированной молекулы в клетки FreeStyle293-F интродуцировали следующие комбинации экспрессионных векторов:
A. Сконструированная молекула: GPC3 ERY5
Полипептиды, кодируемые полинуклеотидами, встроенными в экспрессионные векторы: GPC3 ERY5_Hh (SEQ ID NO: 36; зрелая последовательность, которая не содержит 19 аминокислот на аминоконце, служащих в качестве сигнальной последовательности) и GPC3 ERY2_Hk.
Б. Сконструированная молекула: GPC3 ERY6
Полипептиды, кодируемые полинуклеотидами, встроенными в экспрессионные векторы: GPC3 ERY6_Hk (SEQ ID NO: 37; зрелая последовательность, которая не содержит 19 аминокислот на аминоконце, служащих в качестве сигнальной последовательности) и GPC3 ERY6 Hh (SEQ ID NO: 38; зрелая последовательность, которая не содержит 19 аминокислот на аминоконце, служащих в качестве сигнальной последовательности).
B. Сконструированная молекула: GPC3 ERY7
Полипептиды, кодируемые полинуклеотидами, встроенными в экспрессионные векторы: GPC3 ERY7 Hh (SEQ ID NO: 39; зрелая последовательность, которая не содержит 19 аминокислот на аминоконце, служащих в качестве сигнальной последовательности), GPC3 ERY7 L (SEQ ID NO: 40; зрелая последовательность, которая не содержит 19 аминокислот на аминоконце, служащих в качестве сигнальной последовательности) и GPC3 ERY2_Hk.
Полученный супернатант культуры вносили в колонку Anti FLAG М2 (фирма Sigma). После отмывки колонку элюировали FLAG-пептидом в концентрации 0,1 мг/мл (фирма Sigma). Фракцию, содержащую созданную молекулу, вносили в колонку HisTrap HP (фирма GE Healthcare). После отмывки колонку элюировали концентрационным градиентом имидазола. Фракцию, содержащую созданную молекулу, концентрировали путем ультрафильтрации. Затем фракцию вносили в колонку Супердекс 200 (фирма GE Healthcare). Из элюата собирали только мономерную фракцию, получая каждую очищенную созданную молекулу.
Эти полипептидные комплексы сравнивали с GPC3 BiTE с точки зрения цитотоксической активности. В результате продемонстрировано, что цитотоксическая активность этих полипептидных комплексов оказалась не столь высокой, как у GPC3 BiTE (фиг.2-4). Этот результат позволяет предположить, что добавление Fc к структуре BiTE или имитирующей указанную конструкцию структуре и наличие конфигурация, которая позволяет осуществлять двухвалентное связывание с раковым антигеном, не приводит к созданию требуемой молекулы.
Пример 3. Конструирование и оценка GPC3 ERY8-2. GPC3 ERY9-1 и GPC3 ERY10-1
(1) Конструирование GPC3 ERY8-2. GPC3 ERY9-1 и GPC3 ERY10-1
Далее создавали молекулы, не имеющие структуру BiTE, но обладающие требуемой активностью. В качестве основы применяли IgG к раковому антигену (GPC3) и создавали молекулу, в которой к этому каркасу добавляли scFv к эпсилон-цепи CD3. Fc IgG, применяемый в качестве каркаса, как и в описанных выше случаях, представлял собой молчащий Fc с пониженной активностью связывания с FcgR (Fcγ-рецептор). Конструировали GPC3 ERY8-2 (фиг.17Ж), GPC3 ERY10-1 (фиг.17И) и GPC3 ERY9-1 (фиг.173), в которых scFv к эпсилон-цепи CD3 присоединяли к N-концу Н-цепи, С-концу Н-цепи и С-концу L-цепи антитела к GPC3 IgG-типа соответственно.
Конкретно, применяли известный специалистам в данной области метод, такой как ПЦР с использованием праймеров, содержащих такие же соответствующие дополнительные последовательности, что и в описанном выше методе, для получения серий экспрессионных векторов, в которые встраивали полинуклеотид, кодирующий GPC3 ERY8-2_Hk (SEQ ID NO: 41; зрелая последовательность, которая не содержит 19 аминокислот на аминоконце, служащих в качестве сигнальной последовательности), GPC3 ERY8-2 Hh (SEQ ID NO: 42; зрелая последовательность, которая не содержит 19 аминокислот на аминоконце, служащих в качестве сигнальной последовательности), GPC3 ERY9-1H (SEQ ID NO: 43; зрелая последовательность, которая не содержит 19 аминокислот на аминоконце, служащих в качестве сигнальной последовательности), GPC3 ERY9-1_L-His (SEQ ID NO: 44; зрелая последовательность, которая не содержит 19 аминокислот на аминоконце, служащих в качестве сигнальной последовательности), GPC3 ERY9-1L-FLAG (SEQ ID NO: 45; зрелая последовательность, которая не содержит 19 аминокислот на аминоконце, служащих в качестве сигнальной последовательности) или GPC3 ERY10-1_Hh (SEQ ID NO: 46; зрелая последовательность, которая не содержит 19 аминокислот на аминоконце, служащих в качестве сигнальной последовательности).
Для кратковременной экспрессии каждой созданной молекулы в клетки FreeStyle293-F интродуцировали следующие комбинации экспрессионных векторов.
Г. Сконструированная молекула: GPC3 ERY8-2
Полипептиды, кодируемые полинуклеотидами, встроенными в экспрессионные векторы: GPC3 ERY8-2 Hk (SEQ ID NO: 41; зрелая последовательность, которая не содержит 19 аминокислот на аминоконце, служащих в качестве сигнальной последовательности), GPC3 ERY8-2 Hh (SEQ ID NO: 42; зрелая последовательность, которая не содержит 19 аминокислот на аминоконце, служащих в качестве сигнальной последовательности) и GPC3 ERY7L.
Д. Сконструированная молекула: GPC3 ERY9-1
Полипептиды, кодируемые полинуклеотидами, встроенными в экспрессионные векторы: GPC3 ERY9-1 Н (SEQ ID NO: 43; зрелая последовательность, которая не содержит 19 аминокислот на аминоконце, служащих в качестве сигнальной последовательности), GPC3 ERY9-1_L-His (SEQ ID NO: 44; зрелая последовательность, которая не содержит 19 аминокислот на аминоконце, служащих в качестве сигнальной последовательности) и GPC3 ERY9-1L-FLAG (SEQ ID NO: 45; зрелая последовательность, которая не содержит 19 аминокислот на аминоконце, служащих в качестве сигнальной последовательности). Е. Сконструированная молекула: GPC3 ERY10-1 Полипептиды, кодируемые полинуклеотидами, встроенными в экспрессионные векторы: GPC3 ERY10-1_Hh (SEQ ID NO: 46; зрелая последовательность, которая не содержит 19 аминокислот на аминоконце, служащих в качестве сигнальной последовательности) и GPC3 ERY8-2_Hk, GPC3 ERY7 L.
Полученный супернатант культуры вносили в колонку Anti FLAG М2 (фирма Sigma). После отмывки колонку элюировали FLAG-пептидом в концентрации 0,1 мг/мл (фирма Sigma). Фракцию, содержащую созданную молекулу, вносили в колонку HisTrap HP (фирма GE Healthcare). После отмывки колонку элюировали концентрационным градиентом имидазола. Фракцию, содержащую созданную молекулу, концентрировали путем ультрафильтрации. Затем фракцию вносили в колонку Супердекс 200 (фирма GE Healthcare). Из элюата собирали только мономерную фракцию, получая каждую очищенную созданную молекулу.
Эти молекулы оценивали в отношении цитотоксической активности in vitro. Результат свидетельствует о том, что все молекулы обладали цитотоксической активностью, сопоставимой или более высокой по сравнению с активностью GPC3 BiTE (фиг.5). В частности, установлено, что GPC3 ERY9-1 и GPC3 ERY10-1 обладали выраженно более высокой цитотоксической активностью, чем GPC3 BiTE. В настоящем изобретении впервые продемонстрировано, что молекулы, созданные путем добавления scFv к эпсилон-цепи CD3 к IgG к раковому антигену, обладали цитотоксической активностью, сопоставимой или более высокой, чем у BiTE. В частности, при создании изобретения неожиданно было установлено, что такие молекулы, как GPC3 ERY9-1 и GPC3 ERY10-1, обладали выраженно более высокой цитотоксической активностью по сравнению с BiTE, несмотря на большое расстояние между их связывающим раковый антиген доменом и связывающим доменом эпсилон-цепи CD3.
(2) Оценка эффективности in vivo GPC3 ERY8-2 и GPC3 ERY10-1:
Определяли эффективность in vivo GPC3 ERY8-2 и GPC3 ERY10-1, для которых продемонстрировано наличие цитотоксической активности, сопоставимой или более высокой по сравнению GPC3 BiTE при оценке с использованием анализа in vitro, описанного в разделе (1). Экспрессирующие GPC3 клетки клеточной линии человеческого рака легкого PC-10 смешивали с человеческими РВМС, а затем трансплантировали мышам с тяжелым комбинированным иммунодефицитом линии NOD. Мышей обрабатывали путем введения GPC3 ERY8-2 или GPC3 ERY10-1 (что обозначали как «pre-mix» модель).
В частности, оценку эффективности GPC3 ERY8-2 на «рге-mix» модели с использованием РС-10 осуществляли следующим образом. РВМС выделяли из крови здоровых доноров. NK-клетки выделяли из РВМС с помощью микрогранул CD56 MicroBeads, предназначенных для разделения человеческой крови (фирма MCAS Miltenyi biotec). Смешивали клеточную линию человеческой плоскоклеточной карциномы легкого РС-10 (фирма Immuno-Biological Laboratories Co., Ltd.) (5×106 клеток), человеческие РВМС без NK-клеток (4,5×106 клеток) и (искусственную) базальную мембрану Матригель Матрикс (фирма BD) и затем трансплантировали подкожно в паховую область мышей с тяжелым комбинированным иммунодефицитом линии NOD (фирма CLEA Japan Inc.; самки возрастом 7 недель). День трансплантации обозначали как день 0. За 1 день до трансплантации мышам внутрибрюшинно вводили антитело к асиало-ганглиозиду M1 (асиало-GM1) (фирма Wako Pure Chemical Industries) из расчета 0,2 мг/особь. Через 2 ч после трансплантации вводили внутрибрюшинно GPC ERY8-2 из расчета 30 мкг/особь. GPC ERY8-2 вводили в целом пять раз в течение периода времени с 0 по 4 день.
Кроме того, оценку эффективности GPC3 ERY10-1 на «рге-mix» модели с использованием РС-10 осуществляли следующим образом. РВМС выделяли из крови здоровых доноров. NK-клетки выделяли из РВМС с помощью микрогранул CD56 MicroBeads, предназначенных для разделения человеческой крови (фирма MCAS Miltenyi biotec). Смешивали клеточную линию человеческой плоскоклеточной карциномы легкого РС-10 (фирма Immuno-Biological Laboratories Co., Ltd.) (5×106 клеток), человеческие РВМС без NK-клеток (4,5×106 клеток) и базальную мембрану Матригель Матрикс (фирма BD) и затем трансплантировали подкожно в паховую область мышей с тяжелым комбинированным иммунодефицитом линии NOD (фирма CLEA Japan Inc.; самки возрастом 7 недель). День трансплантации обозначали как день 0. За 1 день до трансплантации мышам внутрибрюшинно вводили антитело к асиало-GMl (фирма Wako Pure Chemical Industries) из расчета 0,2 мг/особь. Через 2 ч после трансплантация вводили внутрибрюшинно GPC ERY10-1 из расчета 30 мкг/особь. GPC ERY10-1 вводили в целом 13 раз в течение периодов времени с 0 по 4 день, с 7 по 11 день и с 14 по 16 день.
Результат демонстрирует, что в группах, которых обрабатывали GPC3 ERY8-2 и GPC3 ERY10-1, рост опухолей выраженно подавлялся по сравнению с группой, обработанной растворителем (ЗФР) (фиг.6 и 7).
Кроме того, эффективность in vivo GPC3 ERY10-1 оценивали также с использованием альтернативной модели. В частности, выращивали Т-клетки путем культивирования человеческих РВМС in vitro и затем интродуцировали мышам с тяжелым комбинированным иммунодефицитом линии NOD, которые имели развитые опухоли, образовавшиеся после трансплантации PC-10. Мышей обрабатывали путем введения GPC3 ERY10-1 (обозначена как модель Т-клеточного переноса).
В частности, оценку эффективности GPC3 ERY10-1 на основе модели Т-клеточного переноса с использованием клеточной линии PC-10, осуществляли следующим образом. Осуществляли культуральную экспансию Т-клеток с использованием набора для активации/экспансии человеческих клеток (фирма MACS Miltenyi biotec) и РВМС, выделенных из крови здоровых добровольцев. Клеточную линию человеческой плоскоклеточной карциномы легкого PC-10 (фирма Immuno-Biological Laboratories Co., Ltd.) (1×107 клеток) смешивали с базальной мембраной Матригель Матрикс (фирма BD) и затем трансплантировали подкожно в паховую область мышей с тяжелым комбинированным иммунодефицитом линии NOD (фирма CLEA Japan Inc.; самки возрастом 7 недель). День трансплантации обозначали как день 0. За 1 день до трансплантации и в дни 6, 8, 12, 16 и 20 мышам внутрибрюшинно вводили антитело к асиало-GMl (фирма Wako Pure Chemical Industries) из расчета 0,2 мг/особь. В день 6 после трансплантации мышей группировали по размеру опухоли и весу тела, а затем трансплантировали Т-клетки, полученные путем культуральной экспансии, как описано выше, из расчета 1×107 клеток/особь в брюшную полость. Через 2 ч после трансплантации вводили внутрибрюшинно GPC ERY10-1 из расчета 30 мкг/особь. GPC ERY10-1 вводили в общей сложности 5 раз в дни 7, 8, 12, 16 и 17.
Результат свидетельствует о том, что при использовании указанной модели в группе, которую обрабатывали GPC3 ERY10-1, также имело место выраженное противоопухолевое действие по сравнению с группой, обработанной растворителем (ЗФР) (фиг.8).
Описанные выше данные демонстрируют, что серии молекул, в которых к IgG-каркасу, имеющему молчащий Fc, добавлен один scFv антитела к эпсилон-цепи CD3, обладают выраженным противоопухолевым действием in vivo.
(3) Оценка удерживания (ретенции) в плазме
Для решения вопроса о том, обладают ли такие молекулы, как GPC3 ERY8-2, GPC3 ERY9-1 и GPC3 ERY10-1, существенно более продолжительным временем полужизни в плазме по сравнению с GPC3 BiTE, вводили GPC3 ERY9-1 и GPC3 ERY10-1 из расчета 30 мкг/особь мышам с тяжелым комбинированным иммунодефицитом линии NOD, которым не были трансплантированы раковые клетки, и в течение времени определяли их концентрации в плазме.
В частности, осуществляли фармакокинетический (ФК) анализ следующим образом. GPC3 ERY9-1 и GPC3 ERY10-1 вводили внутрибрюшинно мышам с тяжелым комбинированным иммунодефицитом линии NOD NOD (фирма CLEA Japan Inc.; самки возрастом 8 недель) из расчета 30 мкг/особь. Собирали образцы крови из щечной вены мышей с использованием гематокритного капилляра (фирма Terumo) через 15 мин, 2 ч, 1 день, 2 дня и 7 дней после введения. Из крови получали плазму.
GPC3 ERY9-1 и GPC3 ERY10-1 разводили соответствующим образом и добавляли к экспрессирующим GPC3 клеткам линии Ba/F3 (GPC3/BaF) или экспрессирующим эпсилон-цепь человеческого CD3 клеткам линии Ba/F3 (CD3/BaF), давая прореагировать GPC3 ERY9-1 или GPC3 ERY10-1 с GPC3/BaF и CD3/BaF. После отмывки указанных клеток добавляли для дополнительного взаимодействия меченное ФИТЦ вторичное антитело. После отмывки указанных клеток оценивали интенсивность флуоресценции метки на клетках с помощью проточного цитометра Epics XL (фирма Beckman Coulter), получая калибровочную кривую для каждого антитела.
В течение определенного промежутка времени собирали образцы крови мышей, которые были обработаны GPC3 ERY9-1 или GPC3 ERY10-1. Из крови получали плазму и соответствующим образом разводили. С помощью метода, аналогичного описанному выше, который применяли для получения калибровочных кривых, образцы плазмы подвергали взаимодействию с GPC3/BaF или CD3/BaF, определяя количество GPC3 ERY9-1 и GPC3 ERY10-1 в плазме, связанных с каждой клеткой. На основе измеренных величин рассчитывали концентрацию каждого антитела в плазме и строили калибровочные кривые согласно описанному выше методу.
Результат свидетельствует о том, что концентрация как GPC3 ERY9-1, так и GPC3 ERY10-1, в крови сохранялась на уровне, превышающем 10 нМ, через 2 дня после введения (фиг.9 и 10). Эти данные свидетельствуют о том, что такие молекулы, как GPC3 ERY9-1 и GPC3 ERY10-1, обладают значительно более продолжительным временем полужизни в плазме по сравнению с BiTE.
(4) Воздействие молчащего Fc на независимую от ракового антигена индукцию цитокинов
(4-1) Конструирование GPC3 ERY15-1. имеющего FcgR-связывающий Fc
Для решения вопроса о том, могут ли молекулы, такие как GPC3 ERY8-2, GPC3 ERY9-1 и GPC3 ERY10-1, вызывать независимую от ракового антигена индукцию цитокинов, конструировали GPC3 ERY15-1, имеющий FcgR-связывающий Fc (фиг.17К).
В частности, аналогично описанному выше методу, применяли ПЦР с использованием праймеров, содержащих соответствующие дополнительные последовательности, и метод, хорошо известный специалистам в данной области, такой как метод с использованием набора для сайтнаправленного мутагенеза QuikChange Site-Directed Mutagenesis (фирма Stratagene), для создания экспрессионных векторов, в которые встроен полинуклеотид, кодирующий GPC3 ERY15-1_Hh (SEQ ID NO: 47; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности) или GPC3 ERY15-1_Hk (SEQ ID NO: 48; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности).
Экспрессионные векторы для GPC3 ERY15-1_Hh (SEQ ID NO: 47; зрелая последовательность, которая не содержит 19 аминокислот на аминоконце, служащих в качестве сигнальной последовательности), GPC3 ERY15-1_Hk (SEQ ID NO: 48; зрелая последовательность, которая не содержит 19 аминокислот на аминоконце, служащих в качестве сигнальной последовательности) и GPC3 ERY7L совместно интродуцировали в клетки FreeStyle293-F для кратковременной экспрессии GPC3 ERY15-1. Полученный супернатант культуры вносили в колонку Anti FLAG М2 (фирма Sigma). После отмывки колонку элюировали FLAG-пептидом в концентрации 0,1 мг/мл (фирма Sigma). Фракцию, содержащую GPC3 ERY15-1, вносили в колонку HisTrap HP (фирма GE Healthcare). После отмывки колонку элюировали концентрационным градиентом имидазола. Фракцию, содержащую созданную молекулу, концентрировали путем ультрафильтрации. Затем фракцию, содержащую GPC3 ERY15-1, вносили в колонку Супердекс 200 (фирма GE Healthcare). Из элюата собирали только мономерную фракцию GPC3 ERY15-1, получая очищенную молекулу GPC3 ERY15-1.
(4-2) Анализ способности вызывать независимую от ракового антигена индукцию цитокинов
Способность GPC3 ERY15-1 вызывать независимую от ракового антигена индукцию цитокинов сравнивали со способностью GPC3 BiTE, GPC3 ERY9-1, GPC3 ERY10-1 и катумаксомаба. С помощью описанного выше метода получали РВМС из крови здоровых добровольцев. По 50 мкл каждого антитела, концентрацию которого доводили до 40нМ, добавляли к 50 мкл суспензии человеческих РВМС (2 х Ю5 клеток/лунку), а затем к ним добавляли по 100 мкл 10% FBS/D-MEM. Реакционную смесь инкубировали в атмосфере, содержащей 5% газообразного диоксида азота, при 37°С.После инкубации в течение 72 ч собирали супернатант культуры и количественно оценивали цитокины, которые секретировались в супернатант культуры, на основе технологии Cytometric Beads Array (СВА)) с использованием набора человеческих Th1/Th2/Thl7 (фирма BD). Анализ осуществляли в трех повторностях с помощью метода, указанного в прилагаемом протоколе.
Установлено, что GPC3 ERY15-1 и катумаксомаб, которые имеют FcgR-связывающий Fc, обладали выраженной способностью индуцировать цитокины. В противоположность этому, у антитела GPC3 BiTE, у которого отсутствовал Fc, и у GPC3 ERY9-1 и GPC3 ERY10-1, которые несли молчащий Fc, способность индуцировать цитокины не была обнаружена (фиг.11). Этот результат позволяет предположить, что молекулы, которые имеют молчащий Fc, такие как GPC3 ERY8-2, GPC3 ERY9-1 и GPC3 ERY10-1, представляют собой отличающиеся высокой безопасностью молекулы, которые не обладают способностью индуцировать цитокины независимым от ракового антигена образом.
Пример 4. Конструирование и оценка GPC3 ERY18 L1, L2, L3, L4 и S1
Оценивали молекулы, имеющие CD3-связывающий домен, отличный по структуре от scFv. Конструировали GPC3 ERY18 (Fig. 17Л), в котором VH- и VL-домены антитела к CD3 были соединены с С-концами двух Н-цепей IgG к раковому антигену GPC3. С помощью такого конструирования создавали серии молекул (GPC3 ERY18 LI, L2, L3 и L4), которые имели в области стыка 1-4 линкерных единицы (Gly-Gly-Gly-Gly-Ser). Одновременно конструировали другую молекулу (GPC3 ERY18 S1), в которой аминокислоты в соответствующих положениях были заменены на Cys, что позволяло интродуцировать дисульфидный мостик.
С частности, осуществляли метод, известный в данной области, такой как ПЦР с использованием праймеров, содержащих, как и в описанном выше методе, соответствующие дополнительные последовательности, для создания серий экспрессионных векторов, в которые встраивали полинуклеотид, кодирующий GPC3 ERY18 L1_Hh (SEQ ID NO: 49; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности), GPC3 ERY18 LIHk (SEQ ID NO: 50; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности), GPC3 ERY18 L2_Hh (SEQ ID NO: 51; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности), GPC3 ERY18 L2_Hk (SEQ ID NO:52; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности), GPC3 ERY18 L3_Hh (SEQ ID NO: 53; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности), GPC3 ERY18 L3_Hk (SEQ ID NO: 54; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности), GPC3 ERY18 L4_Hh (SEQ ID NO: 55; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности), GPC3 ERY18 L4_Hk (SEQ ID NO: 56; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности), GPC3 ERY18 SIHh (SEQ ID NO: 57; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности) или GPC3 ERY18 SIHk (SEQ ID NO: 58; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности).
Для кратковременной экспрессии сконструированных молекул в клетки FreeStyle293-F интродуцировали следующие комбинации экспрессионных векторов.
Ж. Сконструированная молекула: GPC3 ERY18 L1
Экспрессионный векторы: GPC3 ERY18 LI Hh (SEQ ID NO: 49; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности), GPC3 ERY18 LIHk (SEQ ID NO: 50; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности) и GPC3 ERY7 L.
3. Сконструированная молекула: GPC3 ERY18 L2
Экспрессионные векторы: GPC3 ERY18 L2_Hh (SEQ ID NO: 51; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности), GPC3 ERY18 L2 Нк (SEQ ID NO: 52; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности) и GPC3 ERY7 L.
И. Сконструированная молекула: GPC3 ERY18 L3
Экспрессионный векторы: GPC3 ERY18 L3_Hh (SEQ ID NO: 53; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности), GPC3 ERY18 L3_Hk (SEQ ID NO: 54; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности) и GPC3 ERY7 L.
К. Сконструированная молекула: GPC3 ERY18 L4
Экспрессионные векторы: GPC3 ERY18 L4_Hh (SEQ ID NO: 55; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности), GPC3 ERY18 L4_Hk (SEQ ID NO: 56; зрелая последовательность, которая не содержит на аминоконце
19 аминокислот, служащих в качестве сигнальной последовательности) и GPC3 ERY7 L.
Л. Сконструированная молекула: GPC3 ERY18 S1
Экспрессионные векторы: GPC3 ERY18 SI Hh (SEQ ID NO: 57; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности), GPC3 ERY18 SIHk (SEQ ID NO: 58; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности) и GPC3 ERY7 L.
Полученный супернатант культуры вносили в колонку Anti FLAG М2 (фирма Sigma). После отмывки колонку элюировали FLAG-пептидом в концентрации 0,1 мг/мл (фирма Sigma). Фракцию, содержащую созданную молекулу, вносили в колонку HisTrap HP (фирма GE Healthcare). После отмывки колонку элюировали концентрационным градиентом имидазола. Фракцию, содержащую созданную молекулу, концентрировали путем ультрафильтрации. Затем фракцию вносили в колонку Супердекс 200 (фирма GE Healthcare). Из элюата собирали только мономерную фракцию, получая каждую очищенную созданную молекулу.
Для каждой из молекул GPC3 ERY18 LI, GPC3 ERY18L2, GPC3 ERY18L3, GPC3 ERY18L4 и GPC3 ERY18S1 оценивали in vitro цитотоксическую активность (фиг.12 и 13). Результат свидетельствует о том, что все молекулы за исключением GPC3 ERY18 L1 имели активность, сопоставимую с активностью GPC3 ERY10-1. Этот результат демонстрирует, что молекулы, которые имеют структуру, отличную от структуры scFv, обладают сопоставимой цитотоксической активностью. Структура, в которой и VH- и VL-домены антитела к CD3 сцеплены с С-концами двух Н-цепей IgG к раковому антигену (GPC3), вероятно, принимает участие в стабилизации полипептидных комплексов, предлагаемых в настоящем изобретении.
Пример 5. Конструирование и оценка GPC3 ERY19-3
Затем исследовали молекулы, имеющие Fab-подобный CD3-связывающий домен. Конструировали GPC3 ERY19-3 (фиг.17М), в котором VH- и CH1-домены и VL- и CL-домены антитела к CD3 все были связаны с С-концами двух Н- цепей антитела к противораковому антигену (GPC3) IgG-типа. В частности, осуществляли метод, известный в данной области, такой как ПЦР с использованием праймеров, содержащих, как и в описанном выше методе, соответствующие дополнительные последовательности, создавая экспрессионные векторы, в которые встраивали полинуклеотид, кодирующий GPC3 ERY19-3 Hh (SEQ ID NO: 59; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности) или GPC3 ERY19-3_Hk (SEQ ID NO: 60; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности).
Экспрессионные векторы для GPC3 ERY19-3 Hh (SEQ ID NO: 59; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности), GPC3 ERY19-3 Hk (SEQ ID NO: 60; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности) и GPC3 ERY7L совместно интродуцировали в клетки FreeStyle293-F для кратковременной экспрессии GPC3 ERY19-3. Полученный супернатант культуры вносили в колонку с рекомбинантным белком A HiTrap rProtein A FF (фирма GE Healthcare). После отмывки колонку элюировали кислотой. Фракцию, содержащую GPC3 ERY19-3, концентрировали путем ультрафильтрации и затем вносили в колонку Супердекс 200 (фирма GE Healthcare). Из элюата собирали только мономерную фракцию GPC3 ERY19-3, получая очищенную молекулу GPC3 ERY19-3.
Оценивали цитотоксическую активность молекулы GPC3 ERY19-3 in vitro. Результат продемонстрировал, что молекула обладала активностью, сопоставимой с активностью GPC3 BiTE (фиг.14). CD3-связывающий домен с Fab-подобной структурой, по-видимому, принимает участие в стабилизации молекул полипептидных комплексов, предлагаемых в настоящем изобретении.
Пример 6. Получение полипептидных комплексов с использованием только стадии очистки на белке А путем интродукции мутации в СН3-домен GPC3 ERY 10-1
(1) Основные принципы
В GPC3 ERY10-1, полученном согласно методу, описанному в примере 3, СН3-домен имеет структуру типа «выступ-впадина». Требуемую молекулу GPC3 ERY10-1, в которой две Н-цепи гетеромерно ассоциированы, очищали с помощью двух типов очистки на основе аффинности с использованием His-метки и FLAG-метки, присоединенных к С-концу каждой Н-цепи. Если молекулу GPC3 ERY10-1 получают в качестве фармацевтического агента, то сначала осуществляют хроматографию на белке А супернатанта культуры экспрессирующих GPC3 ERY10-1 клеток для очистки полипептидного комплекса, имеющего Fc-домен. За этой стадией следует дополнительная стадия хроматографической очистки с использованием аффинной хроматографии на основе His-метки и аффинной хроматографии на основе FLAG-метки. Это повышает стоимость процесса очистки. Таким образом, в приведенном ниже примере исследовали молекулярные модификации, которые позволяют очищать требуемую молекулу GPC3 ERY10-1, имеющую две гетеромерно ассоциированные Н-цепи, с помощью только хроматографии на белке А без использования His-метки и FLAG-метки.
В частности, исследовали модификации, приводящие к элиминации связывания белка А в одной из двух Н-цепей. В результате таких модификаций, при которых имела место гомомерная ассоциация не связывающих белок А Н-цепей, молекула не могла связываться с белком А и поэтому проходила через колонку с белком А при хроматографии. С другой стороны, молекулу в которой имела место гетеромерная ассоциация не связывающей белок А Н-цепи со связывающей белок А Н-цепью, и молекулу, в которой имела место гомомерная ассоциация связывающих белок А Н-цепей, можно разделять с помощью хроматографии на белке А на основе различий в аффинности к белку А. Однако в Fc-домене антитела сайт связывания белка А перекрывается с сайтом связывания FcRn, что имеет решающее значение для времени удерживания антитела в плазме. Таким образом, необходимо избирательно снижать только активность связывания с белком А, поддерживая при этом активность связывания с FcRn. В качестве такой модификации была выявлена замена His в положении 435 (EU-нумерация) на Arg. Комбинацию указанной мутации с мутациями, описанными в WO 2006/106905 (замена Asp в положении 356 (EU-нумерация) в одной из Н-цепей на Lys и Lys в положении 439 (EU-нумерация) в другой Н-цепи на Glu), которые усиливают гетеромерную ассоциацию двух Н-цепей, оценивали в отношении того, может ли это облегчать очистку полипептидных комплексов, таких как GPC3 ERY10-1, с использованием только хроматографии на белке А.
(2) Конструирование экспрессионных векторов, несущих гены антитела, и экспрессия соответствующих антител
Для получения вариабельной области Н-цепи антитела конструировали ген, кодирующий GC33(2)H (вариабельная область Н-цепи антитела к человеческому глипикану, SEQ ID NO: 61; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности), с помощью метода, известного специалистам в данной области. Аналогично этому, для получения L-цепи антитела конструировали ген, кодирующий GC33-k0 (L-цепь антитела к человеческому глипикану, SEQ ID NO: 62; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности), с помощью метода, известного специалистам в данной области. Затем для получения константной области Н-цепи антитела конструировали описанные ниже гены с помощью метода, известного специалистам в данной области.
М. Сконструированная молекула: LALA-Gld
LALA-Gld (SEQ ID NO: 63; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности), в которой Leu в положениях 234 и 235 (EU-нумерация) заменены на Ala, Asn в положении 297 заменен на Ala, a Gly и Lys на С-конце удалены из последовательности IgG1.
Н. Сконструированная молекула: LALA-Gld-CD3
LALA-Gld-CD3 (SEQ ID NO: 64; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности), в которой CD3 scFv (вариабельная область Н-цепи антитела к человеческому CD3 и вариабельная область L-цепи антитела к человеческому CD3 соединены через полипептидный линкер) сцеплены с С-концом LALA-Gld (SEQ ID NO: 63; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности).
О. Сконструированная молекула: LALA-G3S3E-G1d
LALA-G3S3E-Gld (SEQ ID NO: 65; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности), в которой His в положении 435 (EU-нумерация) заменен на Arg и Lys в положении 439 (EU-нумерация) заменен на Glu в последовательности LALA-G1d (SEQ ID NO: 63; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности).
П. Сконструированная молекула: LALA-S3K-G1d-CD3
LALA-S3K-G1d-CD3 (SEQ ID NO: 66; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности), в которой Asp в положении 356 (EU- нумерация) заменен на Lys в последовательности LALA-G1d-CD3 (SEQ ID NO: 64; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности).
Гены NTA1L и NTA1R Н-цепей антитела к человеческому GPC3 конструировали, присоединяя LALA-G1d-CD3 или LALA-G1d по ходу транскрипции относительно GC33(2)H соответственно. А гены NTA2L и NTA2R Н-цепей антитела к человеческому GPC3 конструировали, присоединяя LALA-S3K-Gld-CD3 или LALA-G3S3E-Gld по ходу транскрипции относительно GC33(2)H соответственно.
Экспрессионные векторы для NTA1L, NTA1R, NTA2L, NTA2R (Н-цепи) и GC33-k0 (L-цепь) конструировали, встраивая каждый ген в экспрессионный вектор, пригодный для клеток животных. Эти векторы объединяли согласно описанному ниже методу и интродуцировали в клетки FreeStyle293 (фирма Invitrogen) с помощью метода, известного специалистам в данной области, для кратковременной экспрессии полипептидных комплексов, описанных ниже. Как указано ниже, полипептидные комплексы обозначали по названиям интродуцированных генов, объединенных в следующем порядке [первая Н-цепь/вторая Н-цепь/Е-цепь].
NTA1L/NTAlR/GC33-k0
NTA2L/NTA2R/GC33-k0
(3) Очистка экспрессированных образцов и оценка их в отношении способности образовывать гетеромерные комплексы Клеточный супернатант FreeStyle293-mieTOK (обозначен далее как СМ), содержащий указанный ниже полипептидный комплекс, применяли в качестве образца.
NTA1L/NTAlR/GC33-k0
NTA2L/NTA2R/GC33-k0
СМ фильтровали через фильтр с диаметром пор 0,22 мкм и вносили в колонку, заполненную rProtein А-Сефарозой Fast Flow (фирма GE Healthcare), уравновешенную с помощью D-ЗФР. Стадии отмывки 1 и 2 и стадию элюции 1 осуществляли с использованием буферов, указанных в таблице 1. Вносимое количество СМ регулировали так, чтобы вносимое количество антитела составляло 20 мг/мл смолы. Элюированные фракции собирали и анализировали с помощью гель-фильтрации для идентификации компонентов во фракциях.

Результаты анализа методом гель-фильтрации каждой элюированной фракции представлены на фиг.15 и в таблице 2. Указанные величины представляют собой площадь элюированного пика в процентах. При экспрессии NTA1L/NTA1R/GC33-k0 или NTA2L/NTA2R/GC33-k0 гомомерное антитело к GPC3 (NTA1L/GC33-k0 or NTA2L/GC33-k0) практически не удалось обнаруживать в СМ. При этом, содержание гомомерного антитела к GPC3 (NTA2R/GC33-k0) в СМ составляло лишь примерно 2% при экспрессии NTA2L/NTA2R/GC33-k0, в то время как его содержание в СМ составляло примерно 76% при экспрессии NTA1L/NTAlR/GC33-k0. Этот результат демонстрирует, что, когда His в положении 435 (EU-нумерация) заменяют на Arg, и для обеспечения эффективного образования гетеромерной молекулы, содержащей соответствующие Н-цепи. Asp в положении 356 (EU-нумерация) в полипептидной последовательности одной из Н-цепей заменяют на Lys, a Lys в положении 439 (EU-нумерация) в полипептидной последовательности другой Н-цепи заменяют на Glu, то гетеромерные полипептидные комплексы, имеющие такую же молекулярную форму, что и GPC3 ERY10-1, можно эффективно очищать, достигая степени чистоты более 98%, только с помощью одной стадии очистки с использованием белка А.

Пример 7. Конструирование и оценка GPC3 ERY 17-2 и GPC3 ERY 17-3
(1) Конструирование GPC3 ERY 17-2 и GPC3 ERY 17-3
Затем, создавали молекулу путем использования IgG к противораковому антигену (GPC3) в качестве каркаса и замены одного из Fab на домен, связывающий эпсилон-цепь CD3. Также как и в описанных выше вариантах, Fc применяемого в качестве каркаса IgG представлял собой молчащий Fc с пониженной активностью связывания с FcgR (Fcγ-рецептор). Для доменов, связывающих эпсилон-цепь CD3, изменяли VH- и VL-домены Fab антитела к эпсилон-цепи CD3 для получения GPC3 ERY17-2 (фиг.19А), а СН1- и CL-домены изменяли для получения GPC3 ERY17-3 (фиг.19Б).
В частности, с помощью метода, хорошо известного специалистам в данной области, такого как ПЦР с использованием праймеров, содержащих такие же соответствующие дополнительные последовательности, что и в описанном выше методе, конструировали серии экспрессионных векторов, в которые был встроен полинуклеотид, кодирующий ERY17-2 Hh (SEQ ID NO: 73; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности), ERY17-2L (SEQ ID NO: 74; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности), ERY17-3_Hh (SEQ ID NO: 75; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности) или ERY17-3 L (SEQ ID NO: 76; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности).
Для кратковременной экспрессии каждой созданной молекулы в клетки FreeStyle293-F интродуцировали следующие комбинации экспрессионных векторов.
Р. Сконструированная молекула: GPC3 ERY17-2
Полипептиды, кодируемые полинуклеотидами, встроенными в экспрессионные векторы: GPC3 ERY8-2 Hk, GPC3 ERY7 L, ERY17-2 Hh (SEQ ID NO: 73; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности) и ERY17-2_L (SEQ ID NO: 74; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности).
С. Сконструированная молекула: GPC3 ERY17-3
Полипептиды, кодируемые полинуклеотидами, встроенными в экспрессионные векторы: GPC3 ERY8-2_Hk, GPC3 ERY7L, ERY17-3_Hh (SEQ ID NO: 75; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности) и ERY17-3 L (SEQ ID NO: 76; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности).
(2) Очистка GPC3 ERY 17-2 и GPC3 ERY 17-3
Полученный супернатант культуры вносили в колонку Anti FLAG М2 (фирма Sigma). После отмывки колонку элюировали FLAG-пептидом в концентрации 0,1 мг/мл (фирма Sigma). Фракцию, содержащую созданную молекулу, вносили в колонку HisTrap HP (фирма GE Healthcare). После отмывки колонку элюировали концентрационным градиентом имидазола. Фракцию, содержащую созданную молекулу, концентрировали путем ультрафильтрации. Затем фракцию вносили в колонку Супердекс 200 (фирма GE Healthcare). Из элюата собирали только мономерную фракцию, получая каждую очищенную созданную молекулу.
(3) Цитотоксическая активность GPC3 ERY 17-2 и GPC3 ERY 17-3
Для GPC3 ERY 17-2 и GPC3 ERY 17-3 оценивали цитотоксическую активность in vitro (фиг.20). Результат продемонстрировал, что обе молекулы обладали заметно более высокой цитотоксической активностью, чем GPC3 BiTE. Таким образом, в настоящем изобретении впервые продемонстрировано, что молекулы, в которых IgG к противораковому антигену применяли в виде каркаса и один из Fab заменяли на домен, связывающий эпсилон-цепь CD3, обладали цитотоксической активностью, сопоставимой или более высокой, чем активность BiTE.
(4) Оценка эффективности GPC3 ERY17-2 на основе модели Т-клеточного переноса с использованием PC-10
Для GPC3 ERY17-2, для которого продемонстрировано наличие цитотоксической активности, сопоставимой или более высокой, чем активность GPC3 BiTE, в анализе in vitro, оценивали эффективность in vivo на основе модели Т-клеточного переноса с использованием клеток PC-10. В частности, оценку эффективности GPC3 ERY17-2 на основе модели Т-клеточного переноса с использованием клеток PC-10 осуществляли следующим образом. Осуществляли культуральную экспансию Т-клеток с использованием набора для активации/экспансии человеческих клеток (фирма MACS Miltenyi biotec) и РВМС, выделенных из крови здоровых добровольцев. Клеточную линию человеческой плоскоклеточной карциномы легкого PC-10 (фирма Immuno-Biological Laboratories Co., Ltd.) (1×107 клеток) смешивали с базальной мембраной Матригель Матрикс (фирма BD) и затем трансплантировали подкожно в паховую область мышей с тяжелым комбинированным иммунодефицитом линии NOD (фирма CLEA Japan Inc.; самки возрастом 7 недель). День трансплантации обозначали как день 0. За 1 день до трансплантации и в дни 13, 17, 21 и 25 мышам вводили внутрибрюшинно антитело к асиало-GMl (фирма Wako Pure Chemical Industries) из расчета 0,2 мг/особь. В день 13 после трансплантации мышей группировали по размеру опухоли и весу тела. В день 14 после трансплантации трансплантировали Т-клетки, полученные путем культуральной экспансии, как описано выше, из расчета 3×107 клеток/особь в брюшную полость. Через 2 ч после трансплантации вводили внутривенно GPC ERY17-2 из расчета 30 мкг/особь. GPC ERY10-1 вводили в целом 5 раз в дни 14, 15, 16, 17 и 18.
Результат продемонстрировал, что при использовании этой модели также имело место выраженное противораковое действие в группе, обработанной GPC3 ERY17-2, по сравнению с группой, обработанной растворителем (фиг.21).
Описанные выше данные продемонстрировали, что молекулы, в которых в качестве каркаса использован противораковый IgG и один из Fab заменен на домен, связывающий эпсилон-цепь CD3, обладали выраженным противораковым действием in vivo.
Пример 8. Конструирование и оценка GPC3 ERY17-2-M20
(1) Конструирование GPC3 ERY17-2-M20
Далее конструировали молекулу, сохраняющую требуемую активность даже после изменений в домене, связывающем эпсилон-цепь CD3. Создавали GPC3 ERY17-2-M20 (фиг.19А), в котором была изменена последовательность домена, связывающего эпсилон-цепь CD3. В частности, с использованием в качестве матрицы экспрессионного вектора для антитела к CD3 (М20) с помощью метода, хорошо известного специалистам в данной области, такого как ПЦР с использованием праймеров, содержащих такие же соответствующие дополнительные последовательности, что и в описанном выше методе, получали серии экспрессионных векторов, в которые был встроен полинуклеотид, кодирующий ERY17-2-M20 Hh (SEQ ID NO: 77; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности) или ERY17-2-M20L (SEQ ID NO: 78; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности).
(2) Очистка GPC3 ERY17-2-M20
Экспрессионные векторы для GPC3 ERY8-2 Hk, GPC3 ERY7 L, ERY17-2-M20_Hh (SEQ ID NO: 77) и ERY17-2-M20L (SEQ ID NO: 78) совместно интродуцировали в клетки FreeStyle293-F для кратковременной экспрессии GPC3 ERY17-2-M20. Полученный супернатант культуры фильтровали через фильтр с диаметром пор 0,22 мкм, а затем вносили в уравновешенную колонку, упакованную rProtein А-Сефарозой Fast Flow (фирма GE Healthcare). Очищенный GPC3 ERY17-2-M20 получали после стадий отмывки 1 и 2 и стадии элюции 1 с использованием буферов, указанных в таблице 3.

(3) Цитотоксическая активность GPC3 ERY17-2-M20
Оценивали цитотоксическую активность GPC3 ERY17-2-M20 in vitro и было установлено, что цитотоксическая активность сопоставима с активностью GPC3 ERY17-2 (фиг.22). Эти данные демонстрируют, что даже молекулы с измененной последовательностью домена, связывающего эпсилон-цепь CD3, обладают сопоставимой цитотоксической активностью.
Пример 9. Конструирование и оценка ЕрСАМ ERY17-2 и ЕрСАМ ERY17-3
(1) Конструирование ЕрСАМ ERY17-2 и ЕрСАМ ERY17-3 Далее конструировали молекулы, мишенью которых являлся другой раковый антиген, но которые сохраняли требуемую активность. Получали ЕрСАМ ERY17-2 (фиг.19А), в котором анти-GPC3 Fab в GPC3 ERY17-2 был заменен на анти-ЕрСАМ Fab, и ЕрСАМ ERY17-3 (фиг.19Б), в котором анти-GPC3 Fab в GPC3 ERY17-3 был заменен на анти-ЕрСАМ Fab. В частности, с использованием в качестве матрицы экспрессионного вектора для антитела к ЕрСАМ, с помощью метода, хорошо известного специалистам в данной области, такого как ПЦР с использованием праймеров, содержащих такие же соответствующие дополнительные последовательности, что и в описанном выше методе, конструировали серии экспрессионных векторов, в которые был встроен полинуклеотид, кодирующий ЕрСАМ ERY17 Hk (SEQ ID NO: 79; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности) или ЕрСАМ ERY17 L (SEQ ID NO: 80; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности).
Для кратковременной экспрессии каждой созданной молекулы в клетки FreeStyle293-F интродуцировали следующие комбинации экспрессионных векторов.
Т. Сконструированная молекула: ЕрСАМ ERY17-2 Полипептиды, кодируемые полинуклеотидами, встроенными в экспрессионные векторы: ЕрСАМ ERY17 Hk (SEQ ID NO: 79; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности), ЕрСАМ ERY17L (SEQ ID NO: 80; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности), ERY17-2 Hh и ERY17-2 L
У. Сконструированная молекула: ЕрСАМ ERY17-3
Полипептиды, кодируемые полинуклеотидами, встроенными в экспрессионные векторы: ЕрСАМ ERY17 Нк, ЕрСАМ ERY17 L, ERY17-3 Hh и ERY17-3 L.
(2) Очистка ЕрСАМ ERY17-2 и ЕрСАМ ERY17-3
Полученный супернатант культуры вносили в колонку Anti FLAG М2 (фирма Sigma). После отмывки колонку элюировали FLAG-пептидом в концентрации 0,1 мг/мл (фирма Sigma). Фракцию, содержащую созданную молекулу, вносили в колонку HisTrap HP (фирма GE Healthcare). После отмывки колонку элюировали концентрационным градиентом имидазола. Фракцию, содержащую созданную молекулу, концентрировали путем ультрафильтрации. Затем фракцию вносили в колонку Супердекс 200 (фирма GE Healthcare). Из элюата собирали только мономерную фракцию, получая каждую очищенную созданную молекулу.
(3) Цитотоксическая активность ЕрСАМ ERY17-2 и ЕрСАМ ERY17-3
Для ЕрСАМ ERY17-2 и ЕрСАМ ERY17-3 оценивали цитотоксическую активность in vitro, и было установлено, что обе молекулы обладали сильной цитотоксической активностью (фиг.23). Таким образом, в настоящем изобретении продемонстрировано, что молекулы, в которых в качестве каркаса использован IgG к противораковому антигену и один из Fab заменен на домен, связывающий эпсилон-цепь CD3, обладали цитотоксической активностью, даже при замене типа ракового антигена.
Пример 10. Конструирование и оценка биспецифических антител с модулированной ассоциацией на поверхности раздела CH1/CL
(1) Создание биспецифического антитела
Путем интродукции мутаций в каждый из СН1- и CL-доменов биспецифического антитела и модулирования тем самым ассоциации на поверхности раздела CH1/CL с использованием отталкивания электрических зарядов на поверхности раздела CH1/CL, можно создавать условия для того, чтобы имела место специфическая ассоциация между Н-цепью и L-цепью антитела к GPC3 и между Н-цепью и L-цепью антитела к CD3. Для того, чтобы модулировать ассоциацию на поверхности раздела CH1/CL с использованием отталкивания электрических зарядов, аминокислотные остатки в СН1 Н-цепей или в CL L-цепей заменяли на Lys, который представляет собой положительно заряженную аминокислоту, или на Glu, который представляет собой отрицательно заряженную аминокислоту.
(2) Конструирование экспрессионных векторов, несущих гены антитела, и экспрессия соответствующих антител
Биспецифическое антитело (фиг.24А) создавали путем модуляции ассоциации на поверхности раздела CH1/CL антитела к CD3 М12 (Н-цепь, SEQ ID NO: 81; L-цепь, SEQ ID NO: 82) и антитела к GPC3 GC33(2) (Н-цепь, SEQ ID NO: 83; L-цепь, SEQ ID NO: 84) и дополнительной интродукции в них модификаций типа «выступ-впадина» (KiH) (WO 1996/027011; Ridgway J. В. и др., Protein Engineering 9, 1996, cc. 617-621, Merchant A.M. и др., Nat. Biotechnol. 16,1998, cc. 677-681) для того, чтобы избежать ассоциации Н-цепей друг с другом. Конструировали также контрольное биспецифическое антитело (фиг.24Б), в которое не интродуцировали ни модуляцию ассоциации на поверхности раздела CH1/CL, ни модификации типа «выступ-впадина» (KiH). В частности, с помощью метода, известного специалистам в данной области, конструировали экспрессионные векторы, несущие в виде вставки полинуклеотид, кодирующий М12 TH2h (SEQ ID NO: 85), в котором несколько аминокислот в СН1 Н-цепи антитела М12 (SEQ ID NO: 81) заменены на Lys, или М12 TL17 (SEQ ID NO: 86), в котором несколько аминокислот в CL L-цепи (SEQ ID NO: 82) заменены на Glu. Аналогично этому, с помощью метода, известного специалистам в данной области, конструировали экспрессионные векторы, несущие в виде вставки полинуклеотид, кодирующий GC33(2)_TH13k (SEQ ID NO: 87) или GC33(2)_TH15k (SEQ ID NO: 88), в котором несколько аминокислот в CH1 Н-цепи антитела GC33(2) (SEQ ID NO: 83) заменены на Glu, или GC33(2)_TL16 (SEQ ID NO: 89) или GC33(2)_TL19 (SEQ ID NO: 90), в котором несколько аминокислот в CL L-цепи (SEQ ID NO: 84) заменены на Lys.
Для кратковременной экспрессии каждой созданной молекулы в клетки FreeStyle293-F интродуцировали следующие комбинации экспрессионных векторов.
Ф. Сконструированная молекула: GM1
Экспрессионные векторы: M12_TH2h (SEQ ID NO: 85), M12TL17 (SEQ ID NO: 86), GC33(2) TH13k (SEQ ID NO: 87) и GC33(2) TL16 (SEQ ID NO: 89)
X. Сконструированная: GM2
Экспрессионные векторы: M12_TH2h (SEQ ID NO: 85), M12TL17 (SEQ ID NO: 86), GC33(2)_TH15k (SEQ ID NO: 88) и GC33(2) TL19 (SEQ ID NO: 90) Ц. Сконструированная молекула: GMO
Экспрессионные векторы: Н-цепь М12 (SEQ ID NO: 81), L-цепь M12 (SEQ ID NO: 82), Н-цепь GC33(2) (SEQ ID NO: 83) и L-цепь of GC33(2) (SEQ ID NO: 84).
Из полученного супернатанта клеток очищали антитела с помощью метода, известного специалистам в данной области, с использованием колонки rProtein А Sepharose™ Fast Flow (фирма GE Healthcare).
(3) Цитотоксическая активность GM1. GM2 и GMO
Оценивали цитотоксическую активность полипептидных комплексов GM1, GM2 и GMO in vitro. Результат демонстрирует, что GM1 и GM2 обладали сопоставимой цитотоксической активностью, и эта активность выше, чем у GM0 (фиг.25). Таким образом, в настоящем изобретении продемонстрировано, что комбинация модуляции ассоциации на поверхности раздела CH1/CL и KiH-модификаций позволяет эффективно получать биспецифические антитела.
Пример 11. Конструирование и оценка EGFR ERY17-2
(1) Конструирование EGFR ERY17-2
Кроме того, создавали молекулу, обладающую требуемой активностью, мишенью которой являются другой раковый антиген. Конструировали EGFR ERY 17-2 (фиг.19А) путем замены aHTH-GPC3 Fab антитела GPC3 ERY17-2 на анти-EGFR Fab. В частности, используя в качестве матрицы экспрессионный вектор для антитела к EGFR, с помощью метода, хорошо известного специалистам в данной области, такого как ПЦР с использованием праймеров, содержащих такие же соответствующие дополнительные последовательности, что и в описанном выше методе, конструировали серии экспрессионных векторов, в которые был встроен полинуклеотид, кодирующий EGFR ERY 17 Hk (SEQ ID NO: 91; зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности) или EGFR ERY17L (SEQ ID NO: 92: зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности).
Для кратковременной экспрессии каждой сконструированной молекулы в клетки FreeStyle293-F интродуцировали следующие комбинации экспрессионных векторов.
Ч. Сконструированная молекула: EGFR ERY17-2
Полипептиды, кодируемые полинуклеотидами, встроенными в экспрессионные векторы: EGFR ERY17_Hk (SEQ ID NO: 91: зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности), EGFR ERY17L (SEQ ID NO: 92: зрелая последовательность, которая не содержит на аминоконце 19 аминокислот, служащих в качестве сигнальной последовательности), ERY17-2_Hh и ERY17-2L.
(2) Очистка EGFR ERY17-2
Полученный супернатант культуры вносили в колонку Anti FLAG М2 (фирма Sigma). После отмывки колонку элюировали FLAG-пептидом в концентрации 0,1 мг/мл (фирма Sigma). Фракцию, содержащую созданную молекулу, вносили в колонку HisTrap HP (фирма GE Healthcare). После отмывки колонку элюировали концентрационным градиентом имидазола. Фракцию, содержащую созданную молекулу, концентрировали путем ультрафильтрации. Затем фракцию вносили в колонку Супердекс 200 (фирма GE Healthcare). Из элюата собирали только мономерную фракцию, получая каждую очищенную созданную молекулу.
(3) Цитотоксическая активность EGFR ERY17-2
Для EGFR ERY17-2 оценивали цитотоксическую активность in vitro, и в этом случае была выявлена сильная цитотоксическая активность (фиг.26). Таким образом, в настоящем изобретении продемонстрировано, что молекулы, в которых в качестве каркаса служил IgG к противораковому антигену и один из Fab был заменен на домен, связывающий эпсилон-цепь CD3, обладали цитотоксической активностью, даже в том случае, когда раковый антиген, представляющий собой GPC3 или ЕрСАМ, дополнительно заменяли на другой тип антигена.
Промышленная применимость
В настоящем изобретении предложены новые полипептидные комплексы, которые сохраняют сильную противораковую активность BiTE и обладают продолжительным временем полужизни в крови, а также характеризуются очень высокой безопасностью, что проявляется в отсутствии индукции независимого от ракового антигена цитокинового шторма, или т.п. При замене антигенсвязывающего домена полипептидного комплекса, предлагаемого в настоящем изобретении, терапевтические агенты, которые содержат в качестве действующего вещества полипептидный комплекс для индукции клеточной цитотоксичности, могут направлено воздействовать на различные клетки, включая раковые клетки, и повреждать их. Таким образом, можно лечить или предупреждать различные типы рака. Это позволяет осуществлять требуемое лечение, отличающееся высокой безопасностью и удобством, и снижать физическую нагрузку на пациентов.
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> ЧУГАИ СЕИЯКУ КАБУШИКИ КАИША
<120> ИНДУЦИРУЮЩИЙ ЦИТОТОКСИЧНОСТЬ ТЕРАПЕВТИЧЕСКИЙ АГЕНТ
<130> C1-A1010Y2P
<150> JP 2010-266760
<151> 2010-11-30
<150> JP 2011-121771
<151> 2011-05-31
<150> JP 2011-238818
<151> 2011-10-31
<160> 92
<170> PatentIn версия 3.5
<210> 1
<211> 1812
<212> ДНК
<213> Homo sapiens
<400> 1
atggccggga ccgtgcgcac cgcgtgcttg gtggtggcga tgctgctcag cttggacttc
60
ccgggacagg cgcagccccc gccgccgccg ccggacgcca cctgtcacca agtccgctcc
120
ttcttccaga gactgcagcc cggactcaag tgggtgccag aaactcccgt gccaggatca
180
gatttgcaag tatgtctccc taagggccca acatgctgct caagaaagat ggaagaaaaa
240
taccaactaa cagcacgatt gaacatggaa cagctgcttc agtctgcaag tatggagctc
300
aagttcttaa ttattcagaa tgctgcggtt ttccaagagg cctttgaaat tgttgttcgc
360
catgccaaga actacaccaa tgccatgttc aagaacaact acccaagcct gactccacaa
420
gcttttgagt ttgtgggtga atttttcaca gatgtgtctc tctacatctt gggttctgac
480
atcaatgtag atgacatggt caatgaattg tttgacagcc tgtttccagt catctatacc
540
cagctaatga acccaggcct gcctgattca gccttggaca tcaatgagtg cctccgagga
600
gcaagacgtg acctgaaagt atttgggaat ttccccaagc ttattatgac ccaggtttcc
660
aagtcactgc aagtcactag gatcttcctt caggctctga atcttggaat tgaagtgatc
720
aacacaactg atcacctgaa gttcagtaag gactgtggcc gaatgctcac cagaatgtgg
780
tactgctctt actgccaggg actgatgatg gttaaaccct gtggcggtta ctgcaatgtg
840
gtcatgcaag gctgtatggc aggtgtggtg gagattgaca agtactggag agaatacatt
900
ctgtcccttg aagaacttgt gaatggcatg tacagaatct atgacatgga gaacgtactg
960
cttggtctct tttcaacaat ccatgattct atccagtatg tccagaagaa tgcaggaaag
1020
ctgaccacca ctgaaactga gaagaaaata tggcacttca aatatcctat cttcttcctg
1080
tgtatagggc tagacttaca gattggcaag ttatgtgccc attctcaaca acgccaatat
1140
agatctgctt attatcctga agatctcttt attgacaaga aagtattaaa agttgctcat
1200
gtagaacatg aagaaacctt atccagccga agaagggaac taattcagaa gttgaagtct
1260
ttcatcagct tctatagtgc tttgcctggc tacatctgca gccatagccc tgtggcggaa
1320
aacgacaccc tttgctggaa tggacaagaa ctcgtggaga gatacagcca aaaggcagca
1380
aggaatggaa tgaaaaacca gttcaatctc catgagctga aaatgaaggg ccctgagcca
1440
gtggtcagtc aaattattga caaactgaag cacattaacc agctcctgag aaccatgtct
1500
atgcccaaag gtagagttct ggataaaaac ctggatgagg aagggtttga aagtggagac
1560
tgcggtgatg atgaagatga gtgcattgga ggctctggtg atggaatgat aaaagtgaag
1620
aatcagctcc gcttccttgc agaactggcc tatgatctgg atgtggatga tgcgcctgga
1680
aacagtcagc aggcaactcc gaaggacaac gagataagca cctttcacaa cctcgggaac
1740
gttcattccc cgctgaagct tctcaccagc atggccatct cggtggtgtg cttcttcttc
1800
ctggtgcact ga 1812
<210> 2
<211> 603
<212> PRT
<213> Homo sapiens
<400> 2
Met Ala Gly Thr Val Arg Thr Ala Cys Leu Val Val Ala Met Leu Leu
1 5 10 15
Ser Leu Asp Phe Pro Gly Gln Ala Gln Pro Pro Pro Pro Pro Pro Asp
20 25 30
Ala Thr Cys His Gln Val Arg Ser Phe Phe Gln Arg Leu Gln Pro Gly
35 40 45
Leu Lys Trp Val Pro Glu Thr Pro Val Pro Gly Ser Asp Leu Gln Val
50 55 60
Cys Leu Pro Lys Gly Pro Thr Cys Cys Ser Arg Lys Met Glu Glu Lys
65 70 75 80
Tyr Gln Leu Thr Ala Arg Leu Asn Met Glu Gln Leu Leu Gln Ser Ala
85 90 95
Ser Met Glu Leu Lys Phe Leu Ile Ile Gln Asn Ala Ala Val Phe Gln
100 105 110
Glu Ala Phe Glu Ile Val Val Arg His Ala Lys Asn Tyr Thr Asn Ala
115 120 125
Met Phe Lys Asn Asn Tyr Pro Ser Leu Thr Pro Gln Ala Phe Glu Phe
130 135 140
Val Gly Glu Phe Phe Thr Asp Val Ser Leu Tyr Ile Leu Gly Ser Asp
145 150 155 160
Ile Asn Val Asp Asp Met Val Asn Glu Leu Phe Asp Ser Leu Phe Pro
165 170 175
Val Ile Tyr Thr Gln Leu Met Asn Pro Gly Leu Pro Asp Ser Ala Leu
180 185 190
Asp Ile Asn Glu Cys Leu Arg Gly Ala Arg Arg Asp Leu Lys Val Phe
195 200 205
Gly Asn Phe Pro Lys Leu Ile Met Thr Gln Val Ser Lys Ser Leu Gln
210 215 220
Val Thr Arg Ile Phe Leu Gln Ala Leu Asn Leu Gly Ile Glu Val Ile
225 230 235 240
Asn Thr Thr Asp His Leu Lys Phe Ser Lys Asp Cys Gly Arg Met Leu
245 250 255
Thr Arg Met Trp Tyr Cys Ser Tyr Cys Gln Gly Leu Met Met Val Lys
260 265 270
Pro Cys Gly Gly Tyr Cys Asn Val Val Met Gln Gly Cys Met Ala Gly
275 280 285
Val Val Glu Ile Asp Lys Tyr Trp Arg Glu Tyr Ile Leu Ser Leu Glu
290 295 300
Glu Leu Val Asn Gly Met Tyr Arg Ile Tyr Asp Met Glu Asn Val Leu
305 310 315 320
Leu Gly Leu Phe Ser Thr Ile His Asp Ser Ile Gln Tyr Val Gln Lys
325 330 335
Asn Ala Gly Lys Leu Thr Thr Thr Glu Thr Glu Lys Lys Ile Trp His
340 345 350
Phe Lys Tyr Pro Ile Phe Phe Leu Cys Ile Gly Leu Asp Leu Gln Ile
355 360 365
Gly Lys Leu Cys Ala His Ser Gln Gln Arg Gln Tyr Arg Ser Ala Tyr
370 375 380
Tyr Pro Glu Asp Leu Phe Ile Asp Lys Lys Val Leu Lys Val Ala His
385 390 395 400
Val Glu His Glu Glu Thr Leu Ser Ser Arg Arg Arg Glu Leu Ile Gln
405 410 415
Lys Leu Lys Ser Phe Ile Ser Phe Tyr Ser Ala Leu Pro Gly Tyr Ile
420 425 430
Cys Ser His Ser Pro Val Ala Glu Asn Asp Thr Leu Cys Trp Asn Gly
435 440 445
Gln Glu Leu Val Glu Arg Tyr Ser Gln Lys Ala Ala Arg Asn Gly Met
450 455 460
Lys Asn Gln Phe Asn Leu His Glu Leu Lys Met Lys Gly Pro Glu Pro
465 470 475 480
Val Val Ser Gln Ile Ile Asp Lys Leu Lys His Ile Asn Gln Leu Leu
485 490 495
Arg Thr Met Ser Met Pro Lys Gly Arg Val Leu Asp Lys Asn Leu Asp
500 505 510
Glu Glu Gly Phe Glu Ser Gly Asp Cys Gly Asp Asp Glu Asp Glu Cys
515 520 525
Ile Gly Gly Ser Gly Asp Gly Met Ile Lys Val Lys Asn Gln Leu Arg
530 535 540
Phe Leu Ala Glu Leu Ala Tyr Asp Leu Asp Val Asp Asp Ala Pro Gly
545 550 555 560
Asn Ser Gln Gln Ala Thr Pro Lys Asp Asn Glu Ile Ser Thr Phe His
565 570 575
Asn Leu Gly Asn Val His Ser Pro Leu Lys Leu Leu Thr Ser Met Ala
580 585 590
Ile Ser Val Val Cys Phe Phe Phe Leu Val His
595 600
<210> 3
<211> 945
<212> ДНК
<213> Homo sapiens
<400> 3
atggcgcccc cgcaggtcct cgcgttcggg cttctgcttg ccgcggcgac ggcgactttt
60
gccgcagctc aggaagaatg tgtctgtgaa aactacaagc tggccgtaaa ctgctttgtg
120
aataataatc gtcaatgcca gtgtacttca gttggtgcac aaaatactgt catttgctca
180
aagctggctg ccaaatgttt ggtgatgaag gcagaaatga atggctcaaa acttgggaga
240
agagcaaaac ctgaaggggc cctccagaac aatgatgggc tttatgatcc tgactgcgat
300
gagagcgggc tctttaaggc caagcagtgc aacggcacct ccatgtgctg gtgtgtgaac
360
actgctgggg tcagaagaac agacaaggac actgaaataa cctgctctga gcgagtgaga
420
acctactgga tcatcattga actaaaacac aaagcaagag aaaaacctta tgatagtaaa
480
agtttgcgga ctgcacttca gaaggagatc acaacgcgtt atcaactgga tccaaaattt
540
atcacgagta ttttgtatga gaataatgtt atcactattg atctggttca aaattcttct
600
caaaaaactc agaatgatgt ggacatagct gatgtggctt attattttga aaaagatgtt
660
aaaggtgaat ccttgtttca ttctaagaaa atggacctga cagtaaatgg ggaacaactg
720
gatctggatc ctggtcaaac tttaatttat tatgttgatg aaaaagcacc tgaattctca
780
atgcagggtc taaaagctgg tgttattgct gttattgtgg ttgtggtgat agcagttgtt
840
gctggaattg ttgtgctggt tatttccaga aagaagagaa tggcaaagta tgagaaggct
900
gagataaagg agatgggtga gatgcatagg gaactcaatg cataa 945
<210> 4
<211> 314
<212> PRT
<213> Homo sapiens
<400> 4
Met Ala Pro Pro Gln Val Leu Ala Phe Gly Leu Leu Leu Ala Ala Ala
1 5 10 15
Thr Ala Thr Phe Ala Ala Ala Gln Glu Glu Cys Val Cys Glu Asn Tyr
20 25 30
Lys Leu Ala Val Asn Cys Phe Val Asn Asn Asn Arg Gln Cys Gln Cys
35 40 45
Thr Ser Val Gly Ala Gln Asn Thr Val Ile Cys Ser Lys Leu Ala Ala
50 55 60
Lys Cys Leu Val Met Lys Ala Glu Met Asn Gly Ser Lys Leu Gly Arg
65 70 75 80
Arg Ala Lys Pro Glu Gly Ala Leu Gln Asn Asn Asp Gly Leu Tyr Asp
85 90 95
Pro Asp Cys Asp Glu Ser Gly Leu Phe Lys Ala Lys Gln Cys Asn Gly
100 105 110
Thr Ser Met Cys Trp Cys Val Asn Thr Ala Gly Val Arg Arg Thr Asp
115 120 125
Lys Asp Thr Glu Ile Thr Cys Ser Glu Arg Val Arg Thr Tyr Trp Ile
130 135 140
Ile Ile Glu Leu Lys His Lys Ala Arg Glu Lys Pro Tyr Asp Ser Lys
145 150 155 160
Ser Leu Arg Thr Ala Leu Gln Lys Glu Ile Thr Thr Arg Tyr Gln Leu
165 170 175
Asp Pro Lys Phe Ile Thr Ser Ile Leu Tyr Glu Asn Asn Val Ile Thr
180 185 190
Ile Asp Leu Val Gln Asn Ser Ser Gln Lys Thr Gln Asn Asp Val Asp
195 200 205
Ile Ala Asp Val Ala Tyr Tyr Phe Glu Lys Asp Val Lys Gly Glu Ser
210 215 220
Leu Phe His Ser Lys Lys Met Asp Leu Thr Val Asn Gly Glu Gln Leu
225 230 235 240
Asp Leu Asp Pro Gly Gln Thr Leu Ile Tyr Tyr Val Asp Glu Lys Ala
245 250 255
Pro Glu Phe Ser Met Gln Gly Leu Lys Ala Gly Val Ile Ala Val Ile
260 265 270
Val Val Val Val Ile Ala Val Val Ala Gly Ile Val Val Leu Val Ile
275 280 285
Ser Arg Lys Lys Arg Met Ala Lys Tyr Glu Lys Ala Glu Ile Lys Glu
290 295 300
Met Gly Glu Met His Arg Glu Leu Asn Ala
305 310
<210> 5
<211> 4
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 5
Gly Gly Gly Ser
1
<210> 6
<211> 4
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 6
Ser Gly Gly Gly
1
<210> 7
<211> 5
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 7
Gly Gly Gly Gly Ser
1 5
<210> 8
<211> 5
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 8
Ser Gly Gly Gly Gly
1 5
<210> 9
<211> 6
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 9
Gly Gly Gly Gly Gly Ser
1 5
<210> 10
<211> 6
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 10
Ser Gly Gly Gly Gly Gly
1 5
<210> 11
<211> 7
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 11
Gly Gly Gly Gly Gly Gly Ser
1 5
<210> 12
<211> 7
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 12
Ser Gly Gly Gly Gly Gly Gly
1 5
<210> 13
<211> 1125
<212> ДНК
<213> Homo sapiens
<400> 13
atgtggttct tgacaactct gctcctttgg gttccagttg atgggcaagt ggacaccaca
60
aaggcagtga tcactttgca gcctccatgg gtcagcgtgt tccaagagga aaccgtaacc
120
ttgcactgtg aggtgctcca tctgcctggg agcagctcta cacagtggtt tctcaatggc
180
acagccactc agacctcgac ccccagctac agaatcacct ctgccagtgt caatgacagt
240
ggtgaataca ggtgccagag aggtctctca gggcgaagtg accccataca gctggaaatc
300
cacagaggct ggctactact gcaggtctcc agcagagtct tcacggaagg agaacctctg
360
gccttgaggt gtcatgcgtg gaaggataag ctggtgtaca atgtgcttta ctatcgaaat
420
ggcaaagcct ttaagttttt ccactggaat tctaacctca ccattctgaa aaccaacata
480
agtcacaatg gcacctacca ttgctcaggc atgggaaagc atcgctacac atcagcagga
540
atatctgtca ctgtgaaaga gctatttcca gctccagtgc tgaatgcatc tgtgacatcc
600
ccactcctgg aggggaatct ggtcaccctg agctgtgaaa caaagttgct cttgcagagg
660
cctggtttgc agctttactt ctccttctac atgggcagca agaccctgcg aggcaggaac
720
acatcctctg aataccaaat actaactgct agaagagaag actctgggtt atactggtgc
780
gaggctgcca cagaggatgg aaatgtcctt aagcgcagcc ctgagttgga gcttcaagtg
840
cttggcctcc agttaccaac tcctgtctgg tttcatgtcc ttttctatct ggcagtggga
900
ataatgtttt tagtgaacac tgttctctgg gtgacaatac gtaaagaact gaaaagaaag
960
aaaaagtggg atttagaaat ctctttggat tctggtcatg agaagaaggt aatttccagc
1020
cttcaagaag acagacattt agaagaagag ctgaaatgtc aggaacaaaa agaagaacag
1080
ctgcaggaag gggtgcaccg gaaggagccc cagggggcca cgtag 1125
<210> 14
<211> 374
<212> PRT
<213> Homo sapiens
<400> 14
Met Trp Phe Leu Thr Thr Leu Leu Leu Trp Val Pro Val Asp Gly Gln
1 5 10 15
Val Asp Thr Thr Lys Ala Val Ile Thr Leu Gln Pro Pro Trp Val Ser
20 25 30
Val Phe Gln Glu Glu Thr Val Thr Leu His Cys Glu Val Leu His Leu
35 40 45
Pro Gly Ser Ser Ser Thr Gln Trp Phe Leu Asn Gly Thr Ala Thr Gln
50 55 60
Thr Ser Thr Pro Ser Tyr Arg Ile Thr Ser Ala Ser Val Asn Asp Ser
65 70 75 80
Gly Glu Tyr Arg Cys Gln Arg Gly Leu Ser Gly Arg Ser Asp Pro Ile
85 90 95
Gln Leu Glu Ile His Arg Gly Trp Leu Leu Leu Gln Val Ser Ser Arg
100 105 110
Val Phe Thr Glu Gly Glu Pro Leu Ala Leu Arg Cys His Ala Trp Lys
115 120 125
Asp Lys Leu Val Tyr Asn Val Leu Tyr Tyr Arg Asn Gly Lys Ala Phe
130 135 140
Lys Phe Phe His Trp Asn Ser Asn Leu Thr Ile Leu Lys Thr Asn Ile
145 150 155 160
Ser His Asn Gly Thr Tyr His Cys Ser Gly Met Gly Lys His Arg Tyr
165 170 175
Thr Ser Ala Gly Ile Ser Val Thr Val Lys Glu Leu Phe Pro Ala Pro
180 185 190
Val Leu Asn Ala Ser Val Thr Ser Pro Leu Leu Glu Gly Asn Leu Val
195 200 205
Thr Leu Ser Cys Glu Thr Lys Leu Leu Leu Gln Arg Pro Gly Leu Gln
210 215 220
Leu Tyr Phe Ser Phe Tyr Met Gly Ser Lys Thr Leu Arg Gly Arg Asn
225 230 235 240
Thr Ser Ser Glu Tyr Gln Ile Leu Thr Ala Arg Arg Glu Asp Ser Gly
245 250 255
Leu Tyr Trp Cys Glu Ala Ala Thr Glu Asp Gly Asn Val Leu Lys Arg
260 265 270
Ser Pro Glu Leu Glu Leu Gln Val Leu Gly Leu Gln Leu Pro Thr Pro
275 280 285
Val Trp Phe His Val Leu Phe Tyr Leu Ala Val Gly Ile Met Phe Leu
290 295 300
Val Asn Thr Val Leu Trp Val Thr Ile Arg Lys Glu Leu Lys Arg Lys
305 310 315 320
Lys Lys Trp Asp Leu Glu Ile Ser Leu Asp Ser Gly His Glu Lys Lys
325 330 335
Val Ile Ser Ser Leu Gln Glu Asp Arg His Leu Glu Glu Glu Leu Lys
340 345 350
Cys Gln Glu Gln Lys Glu Glu Gln Leu Gln Glu Gly Val His Arg Lys
355 360 365
Glu Pro Gln Gly Ala Thr
370
<210> 15
<211> 951
<212> ДНК
<213> Homo sapiens
<400> 15
atgactatgg agacccaaat gtctcagaat gtatgtccca gaaacctgtg gctgcttcaa
60
ccattgacag ttttgctgct gctggcttct gcagacagtc aagctgctcc cccaaaggct
120
gtgctgaaac ttgagccccc gtggatcaac gtgctccagg aggactctgt gactctgaca
180
tgccaggggg ctcgcagccc tgagagcgac tccattcagt ggttccacaa tgggaatctc
240
attcccaccc acacgcagcc cagctacagg ttcaaggcca acaacaatga cagcggggag
300
tacacgtgcc agactggcca gaccagcctc agcgaccctg tgcatctgac tgtgctttcc
360
gaatggctgg tgctccagac ccctcacctg gagttccagg agggagaaac catcatgctg
420
aggtgccaca gctggaagga caagcctctg gtcaaggtca cattcttcca gaatggaaaa
480
tcccagaaat tctcccattt ggatcccacc ttctccatcc cacaagcaaa ccacagtcac
540
agtggtgatt accactgcac aggaaacata ggctacacgc tgttctcatc caagcctgtg
600
accatcactg tccaagtgcc cagcatgggc agctcttcac caatgggggt cattgtggct
660
gtggtcattg cgactgctgt agcagccatt gttgctgctg tagtggcctt gatctactgc
720
aggaaaaagc ggatttcagc caattccact gatcctgtga aggctgccca atttgagcca
780
cctggacgtc aaatgattgc catcagaaag agacaacttg aagaaaccaa caatgactat
840
gaaacagctg acggcggcta catgactctg aaccccaggg cacctactga cgatgataaa
900
aacatctacc tgactcttcc tcccaacgac catgtcaaca gtaataacta a 951
<210> 16
<211> 316
<212> PRT
<213> Homo sapiens
<400> 16
Met Thr Met Glu Thr Gln Met Ser Gln Asn Val Cys Pro Arg Asn Leu
1 5 10 15
Trp Leu Leu Gln Pro Leu Thr Val Leu Leu Leu Leu Ala Ser Ala Asp
20 25 30
Ser Gln Ala Ala Pro Pro Lys Ala Val Leu Lys Leu Glu Pro Pro Trp
35 40 45
Ile Asn Val Leu Gln Glu Asp Ser Val Thr Leu Thr Cys Gln Gly Ala
50 55 60
Arg Ser Pro Glu Ser Asp Ser Ile Gln Trp Phe His Asn Gly Asn Leu
65 70 75 80
Ile Pro Thr His Thr Gln Pro Ser Tyr Arg Phe Lys Ala Asn Asn Asn
85 90 95
Asp Ser Gly Glu Tyr Thr Cys Gln Thr Gly Gln Thr Ser Leu Ser Asp
100 105 110
Pro Val His Leu Thr Val Leu Ser Glu Trp Leu Val Leu Gln Thr Pro
115 120 125
His Leu Glu Phe Gln Glu Gly Glu Thr Ile Met Leu Arg Cys His Ser
130 135 140
Trp Lys Asp Lys Pro Leu Val Lys Val Thr Phe Phe Gln Asn Gly Lys
145 150 155 160
Ser Gln Lys Phe Ser His Leu Asp Pro Thr Phe Ser Ile Pro Gln Ala
165 170 175
Asn His Ser His Ser Gly Asp Tyr His Cys Thr Gly Asn Ile Gly Tyr
180 185 190
Thr Leu Phe Ser Ser Lys Pro Val Thr Ile Thr Val Gln Val Pro Ser
195 200 205
Met Gly Ser Ser Ser Pro Met Gly Val Ile Val Ala Val Val Ile Ala
210 215 220
Thr Ala Val Ala Ala Ile Val Ala Ala Val Val Ala Leu Ile Tyr Cys
225 230 235 240
Arg Lys Lys Arg Ile Ser Ala Asn Ser Thr Asp Pro Val Lys Ala Ala
245 250 255
Gln Phe Glu Pro Pro Gly Arg Gln Met Ile Ala Ile Arg Lys Arg Gln
260 265 270
Leu Glu Glu Thr Asn Asn Asp Tyr Glu Thr Ala Asp Gly Gly Tyr Met
275 280 285
Thr Leu Asn Pro Arg Ala Pro Thr Asp Asp Asp Lys Asn Ile Tyr Leu
290 295 300
Thr Leu Pro Pro Asn Asp His Val Asn Ser Asn Asn
305 310 315
<210> 17
<211> 876
<212> ДНК
<213> Homo sapiens
<400> 17
atgggaatcc tgtcattctt acctgtcctt gccactgaga gtgactgggc tgactgcaag
60
tccccccagc cttggggtca tatgcttctg tggacagctg tgctattcct ggctcctgtt
120
gctgggacac ctgcagctcc cccaaaggct gtgctgaaac tcgagcccca gtggatcaac
180
gtgctccagg aggactctgt gactctgaca tgccggggga ctcacagccc tgagagcgac
240
tccattcagt ggttccacaa tgggaatctc attcccaccc acacgcagcc cagctacagg
300
ttcaaggcca acaacaatga cagcggggag tacacgtgcc agactggcca gaccagcctc
360
agcgaccctg tgcatctgac tgtgctttct gagtggctgg tgctccagac ccctcacctg
420
gagttccagg agggagaaac catcgtgctg aggtgccaca gctggaagga caagcctctg
480
gtcaaggtca cattcttcca gaatggaaaa tccaagaaat tttcccgttc ggatcccaac
540
ttctccatcc cacaagcaaa ccacagtcac agtggtgatt accactgcac aggaaacata
600
ggctacacgc tgtactcatc caagcctgtg accatcactg tccaagctcc cagctcttca
660
ccgatgggga tcattgtggc tgtggtcact gggattgctg tagcggccat tgttgctgct
720
gtagtggcct tgatctactg caggaaaaag cggatttcag ccaatcccac taatcctgat
780
gaggctgaca aagttggggc tgagaacaca atcacctatt cacttctcat gcacccggat
840
gctctggaag agcctgatga ccagaaccgt atttag 876
<210> 18
<211> 291
<212> PRT
<213> Homo sapiens
<400> 18
Met Gly Ile Leu Ser Phe Leu Pro Val Leu Ala Thr Glu Ser Asp Trp
1 5 10 15
Ala Asp Cys Lys Ser Pro Gln Pro Trp Gly His Met Leu Leu Trp Thr
20 25 30
Ala Val Leu Phe Leu Ala Pro Val Ala Gly Thr Pro Ala Ala Pro Pro
35 40 45
Lys Ala Val Leu Lys Leu Glu Pro Gln Trp Ile Asn Val Leu Gln Glu
50 55 60
Asp Ser Val Thr Leu Thr Cys Arg Gly Thr His Ser Pro Glu Ser Asp
65 70 75 80
Ser Ile Gln Trp Phe His Asn Gly Asn Leu Ile Pro Thr His Thr Gln
85 90 95
Pro Ser Tyr Arg Phe Lys Ala Asn Asn Asn Asp Ser Gly Glu Tyr Thr
100 105 110
Cys Gln Thr Gly Gln Thr Ser Leu Ser Asp Pro Val His Leu Thr Val
115 120 125
Leu Ser Glu Trp Leu Val Leu Gln Thr Pro His Leu Glu Phe Gln Glu
130 135 140
Gly Glu Thr Ile Val Leu Arg Cys His Ser Trp Lys Asp Lys Pro Leu
145 150 155 160
Val Lys Val Thr Phe Phe Gln Asn Gly Lys Ser Lys Lys Phe Ser Arg
165 170 175
Ser Asp Pro Asn Phe Ser Ile Pro Gln Ala Asn His Ser His Ser Gly
180 185 190
Asp Tyr His Cys Thr Gly Asn Ile Gly Tyr Thr Leu Tyr Ser Ser Lys
195 200 205
Pro Val Thr Ile Thr Val Gln Ala Pro Ser Ser Ser Pro Met Gly Ile
210 215 220
Ile Val Ala Val Val Thr Gly Ile Ala Val Ala Ala Ile Val Ala Ala
225 230 235 240
Val Val Ala Leu Ile Tyr Cys Arg Lys Lys Arg Ile Ser Ala Asn Pro
245 250 255
Thr Asn Pro Asp Glu Ala Asp Lys Val Gly Ala Glu Asn Thr Ile Thr
260 265 270
Tyr Ser Leu Leu Met His Pro Asp Ala Leu Glu Glu Pro Asp Asp Gln
275 280 285
Asn Arg Ile
290
<210> 19
<211> 765
<212> ДНК
<213> Homo sapiens
<400> 19
atgtggcagc tgctcctccc aactgctctg ctacttctag tttcagctgg catgcggact
60
gaagatctcc caaaggctgt ggtgttcctg gagcctcaat ggtacagggt gctcgagaag
120
gacagtgtga ctctgaagtg ccagggagcc tactcccctg aggacaattc cacacagtgg
180
tttcacaatg agagcctcat ctcaagccag gcctcgagct acttcattga cgctgccaca
240
gttgacgaca gtggagagta caggtgccag acaaacctct ccaccctcag tgacccggtg
300
cagctagaag tccatatcgg ctggctgttg ctccaggccc ctcggtgggt gttcaaggag
360
gaagacccta ttcacctgag gtgtcacagc tggaagaaca ctgctctgca taaggtcaca
420
tatttacaga atggcaaagg caggaagtat tttcatcata attctgactt ctacattcca
480
aaagccacac tcaaagacag cggctcctac ttctgcaggg ggcttgttgg gagtaaaaat
540
gtgtcttcag agactgtgaa catcaccatc actcaaggtt tgtcagtgtc aaccatctca
600
tcattctttc cacctgggta ccaagtctct ttctgcttgg tgatggtact cctttttgca
660
gtggacacag gactatattt ctctgtgaag acaaacattc gaagctcaac aagagactgg
720
aaggaccata aatttaaatg gagaaaggac cctcaagaca aatga 765
<210> 20
<211> 254
<212> PRT
<213> Homo sapiens
<400> 20
Met Trp Gln Leu Leu Leu Pro Thr Ala Leu Leu Leu Leu Val Ser Ala
1 5 10 15
Gly Met Arg Thr Glu Asp Leu Pro Lys Ala Val Val Phe Leu Glu Pro
20 25 30
Gln Trp Tyr Arg Val Leu Glu Lys Asp Ser Val Thr Leu Lys Cys Gln
35 40 45
Gly Ala Tyr Ser Pro Glu Asp Asn Ser Thr Gln Trp Phe His Asn Glu
50 55 60
Ser Leu Ile Ser Ser Gln Ala Ser Ser Tyr Phe Ile Asp Ala Ala Thr
65 70 75 80
Val Asp Asp Ser Gly Glu Tyr Arg Cys Gln Thr Asn Leu Ser Thr Leu
85 90 95
Ser Asp Pro Val Gln Leu Glu Val His Ile Gly Trp Leu Leu Leu Gln
100 105 110
Ala Pro Arg Trp Val Phe Lys Glu Glu Asp Pro Ile His Leu Arg Cys
115 120 125
His Ser Trp Lys Asn Thr Ala Leu His Lys Val Thr Tyr Leu Gln Asn
130 135 140
Gly Lys Gly Arg Lys Tyr Phe His His Asn Ser Asp Phe Tyr Ile Pro
145 150 155 160
Lys Ala Thr Leu Lys Asp Ser Gly Ser Tyr Phe Cys Arg Gly Leu Val
165 170 175
Gly Ser Lys Asn Val Ser Ser Glu Thr Val Asn Ile Thr Ile Thr Gln
180 185 190
Gly Leu Ser Val Ser Thr Ile Ser Ser Phe Phe Pro Pro Gly Tyr Gln
195 200 205
Val Ser Phe Cys Leu Val Met Val Leu Leu Phe Ala Val Asp Thr Gly
210 215 220
Leu Tyr Phe Ser Val Lys Thr Asn Ile Arg Ser Ser Thr Arg Asp Trp
225 230 235 240
Lys Asp His Lys Phe Lys Trp Arg Lys Asp Pro Gln Asp Lys
245 250
<210> 21
<211> 702
<212> ДНК
<213> Homo sapiens
<400> 21
atgtggcagc tgctcctccc aactgctctg ctacttctag tttcagctgg catgcggact
60
gaagatctcc caaaggctgt ggtgttcctg gagcctcaat ggtacagcgt gcttgagaag
120
gacagtgtga ctctgaagtg ccagggagcc tactcccctg aggacaattc cacacagtgg
180
tttcacaatg agagcctcat ctcaagccag gcctcgagct acttcattga cgctgccaca
240
gtcaacgaca gtggagagta caggtgccag acaaacctct ccaccctcag tgacccggtg
300
cagctagaag tccatatcgg ctggctgttg ctccaggccc ctcggtgggt gttcaaggag
360
gaagacccta ttcacctgag gtgtcacagc tggaagaaca ctgctctgca taaggtcaca
420
tatttacaga atggcaaaga caggaagtat tttcatcata attctgactt ccacattcca
480
aaagccacac tcaaagatag cggctcctac ttctgcaggg ggcttgttgg gagtaaaaat
540
gtgtcttcag agactgtgaa catcaccatc actcaaggtt tggcagtgtc aaccatctca
600
tcattctctc cacctgggta ccaagtctct ttctgcttgg tgatggtact cctttttgca
660
gtggacacag gactatattt ctctgtgaag acaaacattt ga 702
<210> 22
<211> 233
<212> PRT
<213> Homo sapiens
<400> 22
Met Trp Gln Leu Leu Leu Pro Thr Ala Leu Leu Leu Leu Val Ser Ala
1 5 10 15
Gly Met Arg Thr Glu Asp Leu Pro Lys Ala Val Val Phe Leu Glu Pro
20 25 30
Gln Trp Tyr Ser Val Leu Glu Lys Asp Ser Val Thr Leu Lys Cys Gln
35 40 45
Gly Ala Tyr Ser Pro Glu Asp Asn Ser Thr Gln Trp Phe His Asn Glu
50 55 60
Ser Leu Ile Ser Ser Gln Ala Ser Ser Tyr Phe Ile Asp Ala Ala Thr
65 70 75 80
Val Asn Asp Ser Gly Glu Tyr Arg Cys Gln Thr Asn Leu Ser Thr Leu
85 90 95
Ser Asp Pro Val Gln Leu Glu Val His Ile Gly Trp Leu Leu Leu Gln
100 105 110
Ala Pro Arg Trp Val Phe Lys Glu Glu Asp Pro Ile His Leu Arg Cys
115 120 125
His Ser Trp Lys Asn Thr Ala Leu His Lys Val Thr Tyr Leu Gln Asn
130 135 140
Gly Lys Asp Arg Lys Tyr Phe His His Asn Ser Asp Phe His Ile Pro
145 150 155 160
Lys Ala Thr Leu Lys Asp Ser Gly Ser Tyr Phe Cys Arg Gly Leu Val
165 170 175
Gly Ser Lys Asn Val Ser Ser Glu Thr Val Asn Ile Thr Ile Thr Gln
180 185 190
Gly Leu Ala Val Ser Thr Ile Ser Ser Phe Ser Pro Pro Gly Tyr Gln
195 200 205
Val Ser Phe Cys Leu Val Met Val Leu Leu Phe Ala Val Asp Thr Gly
210 215 220
Leu Tyr Phe Ser Val Lys Thr Asn Ile
225 230
<210> 23
<211> 330
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 23
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys
1 5 10 15
Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr
65 70 75 80
Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys
100 105 110
Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro
115 120 125
Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys
130 135 140
Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp
145 150 155 160
Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu
165 170 175
Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu
180 185 190
His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn
195 200 205
Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly
210 215 220
Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu
225 230 235 240
Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr
245 250 255
Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn
260 265 270
Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe
275 280 285
Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn
290 295 300
Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr
305 310 315 320
Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
325 330
<210> 24
<211> 326
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 24
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg
1 5 10 15
Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Asn Phe Gly Thr Gln Thr
65 70 75 80
Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Thr Val Glu Arg Lys Cys Cys Val Glu Cys Pro Pro Cys Pro Ala Pro
100 105 110
Pro Val Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
115 120 125
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
130 135 140
Val Ser His Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly
145 150 155 160
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn
165 170 175
Ser Thr Phe Arg Val Val Ser Val Leu Thr Val Val His Gln Asp Trp
180 185 190
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro
195 200 205
Ala Pro Ile Glu Lys Thr Ile Ser Lys Thr Lys Gly Gln Pro Arg Glu
210 215 220
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn
225 230 235 240
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
245 250 255
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
260 265 270
Thr Pro Pro Met Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
275 280 285
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
290 295 300
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
305 310 315 320
Ser Leu Ser Pro Gly Lys
325
<210> 25
<211> 377
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 25
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg
1 5 10 15
Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr
65 70 75 80
Tyr Thr Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Arg Val Glu Leu Lys Thr Pro Leu Gly Asp Thr Thr His Thr Cys Pro
100 105 110
Arg Cys Pro Glu Pro Lys Ser Cys Asp Thr Pro Pro Pro Cys Pro Arg
115 120 125
Cys Pro Glu Pro Lys Ser Cys Asp Thr Pro Pro Pro Cys Pro Arg Cys
130 135 140
Pro Glu Pro Lys Ser Cys Asp Thr Pro Pro Pro Cys Pro Arg Cys Pro
145 150 155 160
Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
165 170 175
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
180 185 190
Val Val Asp Val Ser His Glu Asp Pro Glu Val Gln Phe Lys Trp Tyr
195 200 205
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
210 215 220
Gln Tyr Asn Ser Thr Phe Arg Val Val Ser Val Leu Thr Val Leu His
225 230 235 240
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
245 250 255
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Thr Lys Gly Gln
260 265 270
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met
275 280 285
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
290 295 300
Ser Asp Ile Ala Val Glu Trp Glu Ser Ser Gly Gln Pro Glu Asn Asn
305 310 315 320
Tyr Asn Thr Thr Pro Pro Met Leu Asp Ser Asp Gly Ser Phe Phe Leu
325 330 335
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Ile
340 345 350
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn Arg Phe Thr Gln
355 360 365
Lys Ser Leu Ser Leu Ser Pro Gly Lys
370 375
<210> 26
<211> 327
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 26
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg
1 5 10 15
Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Lys Thr
65 70 75 80
Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Arg Val Glu Ser Lys Tyr Gly Pro Pro Cys Pro Ser Cys Pro Ala Pro
100 105 110
Glu Phe Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
115 120 125
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
130 135 140
Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp
145 150 155 160
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe
165 170 175
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
180 185 190
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu
195 200 205
Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
210 215 220
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys
225 230 235 240
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
245 250 255
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
260 265 270
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
275 280 285
Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser
290 295 300
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
305 310 315 320
Leu Ser Leu Ser Leu Gly Lys
325
<210> 27
<211> 549
<212> ДНК
<213> Homo sapiens
<400> 27
atggaacagg ggaagggcct ggctgtcctc atcctggcta tcattcttct tcaaggtact
60
ttggcccagt caatcaaagg aaaccacttg gttaaggtgt atgactatca agaagatggt
120
tcggtacttc tgacttgtga tgcagaagcc aaaaatatca catggtttaa agatgggaag
180
atgatcggct tcctaactga agataaaaaa aaatggaatc tgggaagtaa tgccaaggac
240
cctcgaggga tgtatcagtg taaaggatca cagaacaagt caaaaccact ccaagtgtat
300
tacagaatgt gtcagaactg cattgaacta aatgcagcca ccatatctgg ctttctcttt
360
gctgaaatcg tcagcatttt cgtccttgct gttggggtct acttcattgc tggacaggat
420
ggagttcgcc agtcgagagc ttcagacaag cagactctgt tgcccaatga ccagctctac
480
cagcccctca aggatcgaga agatgaccag tacagccacc ttcaaggaaa ccagttgagg
540
aggaattga 549
<210> 28
<211> 182
<212> PRT
<213> Homo sapiens
<400> 28
Met Glu Gln Gly Lys Gly Leu Ala Val Leu Ile Leu Ala Ile Ile Leu
1 5 10 15
Leu Gln Gly Thr Leu Ala Gln Ser Ile Lys Gly Asn His Leu Val Lys
20 25 30
Val Tyr Asp Tyr Gln Glu Asp Gly Ser Val Leu Leu Thr Cys Asp Ala
35 40 45
Glu Ala Lys Asn Ile Thr Trp Phe Lys Asp Gly Lys Met Ile Gly Phe
50 55 60
Leu Thr Glu Asp Lys Lys Lys Trp Asn Leu Gly Ser Asn Ala Lys Asp
65 70 75 80
Pro Arg Gly Met Tyr Gln Cys Lys Gly Ser Gln Asn Lys Ser Lys Pro
85 90 95
Leu Gln Val Tyr Tyr Arg Met Cys Gln Asn Cys Ile Glu Leu Asn Ala
100 105 110
Ala Thr Ile Ser Gly Phe Leu Phe Ala Glu Ile Val Ser Ile Phe Val
115 120 125
Leu Ala Val Gly Val Tyr Phe Ile Ala Gly Gln Asp Gly Val Arg Gln
130 135 140
Ser Arg Ala Ser Asp Lys Gln Thr Leu Leu Pro Asn Asp Gln Leu Tyr
145 150 155 160
Gln Pro Leu Lys Asp Arg Glu Asp Asp Gln Tyr Ser His Leu Gln Gly
165 170 175
Asn Gln Leu Arg Arg Asn
180
<210> 29
<211> 516
<212> ДНК
<213> Homo sapiens
<400> 29
atggaacata gcacgtttct ctctggcctg gtactggcta cccttctctc gcaagtgagc
60
cccttcaaga tacctataga ggaacttgag gacagagtgt ttgtgaattg caataccagc
120
atcacatggg tagagggaac ggtgggaaca ctgctctcag acattacaag actggacctg
180
ggaaaacgca tcctggaccc acgaggaata tataggtgta atgggacaga tatatacaag
240
gacaaagaat ctaccgtgca agttcattat cgaatgtgcc agagctgtgt ggagctggat
300
ccagccaccg tggctggcat cattgtcact gatgtcattg ccactctgct ccttgctttg
360
ggagtcttct gctttgctgg acatgagact ggaaggctgt ctggggctgc cgacacacaa
420
gctctgttga ggaatgacca ggtctatcag cccctccgag atcgagatga tgctcagtac
480
agccaccttg gaggaaactg ggctcggaac aagtga 516
<210> 30
<211> 171
<212> PRT
<213> Homo sapiens
<400> 30
Met Glu His Ser Thr Phe Leu Ser Gly Leu Val Leu Ala Thr Leu Leu
1 5 10 15
Ser Gln Val Ser Pro Phe Lys Ile Pro Ile Glu Glu Leu Glu Asp Arg
20 25 30
Val Phe Val Asn Cys Asn Thr Ser Ile Thr Trp Val Glu Gly Thr Val
35 40 45
Gly Thr Leu Leu Ser Asp Ile Thr Arg Leu Asp Leu Gly Lys Arg Ile
50 55 60
Leu Asp Pro Arg Gly Ile Tyr Arg Cys Asn Gly Thr Asp Ile Tyr Lys
65 70 75 80
Asp Lys Glu Ser Thr Val Gln Val His Tyr Arg Met Cys Gln Ser Cys
85 90 95
Val Glu Leu Asp Pro Ala Thr Val Ala Gly Ile Ile Val Thr Asp Val
100 105 110
Ile Ala Thr Leu Leu Leu Ala Leu Gly Val Phe Cys Phe Ala Gly His
115 120 125
Glu Thr Gly Arg Leu Ser Gly Ala Ala Asp Thr Gln Ala Leu Leu Arg
130 135 140
Asn Asp Gln Val Tyr Gln Pro Leu Arg Asp Arg Asp Asp Ala Gln Tyr
145 150 155 160
Ser His Leu Gly Gly Asn Trp Ala Arg Asn Lys
165 170
<210> 31
<211> 624
<212> ДНК
<213> Homo sapiens
<400> 31
atgcagtcgg gcactcactg gagagttctg ggcctctgcc tcttatcagt tggcgtttgg
60
gggcaagatg gtaatgaaga aatgggtggt attacacaga caccatataa agtctccatc
120
tctggaacca cagtaatatt gacatgccct cagtatcctg gatctgaaat actatggcaa
180
cacaatgata aaaacatagg cggtgatgag gatgataaaa acataggcag tgatgaggat
240
cacctgtcac tgaaggaatt ttcagaattg gagcaaagtg gttattatgt ctgctacccc
300
agaggaagca aaccagaaga tgcgaacttt tatctctacc tgagggcaag agtgtgtgag
360
aactgcatgg agatggatgt gatgtcggtg gccacaattg tcatagtgga catctgcatc
420
actgggggct tgctgctgct ggtttactac tggagcaaga atagaaaggc caaggccaag
480
cctgtgacac gaggagcggg tgctggcggc aggcaaaggg gacaaaacaa ggagaggcca
540
ccacctgttc ccaacccaga ctatgagccc atccggaaag gccagcggga cctgtattct
600
ggcctgaatc agagacgcat ctga 624
<210> 32
<211> 207
<212> PRT
<213> Homo sapiens
<400> 32
Met Gln Ser Gly Thr His Trp Arg Val Leu Gly Leu Cys Leu Leu Ser
1 5 10 15
Val Gly Val Trp Gly Gln Asp Gly Asn Glu Glu Met Gly Gly Ile Thr
20 25 30
Gln Thr Pro Tyr Lys Val Ser Ile Ser Gly Thr Thr Val Ile Leu Thr
35 40 45
Cys Pro Gln Tyr Pro Gly Ser Glu Ile Leu Trp Gln His Asn Asp Lys
50 55 60
Asn Ile Gly Gly Asp Glu Asp Asp Lys Asn Ile Gly Ser Asp Glu Asp
65 70 75 80
His Leu Ser Leu Lys Glu Phe Ser Glu Leu Glu Gln Ser Gly Tyr Tyr
85 90 95
Val Cys Tyr Pro Arg Gly Ser Lys Pro Glu Asp Ala Asn Phe Tyr Leu
100 105 110
Tyr Leu Arg Ala Arg Val Cys Glu Asn Cys Met Glu Met Asp Val Met
115 120 125
Ser Val Ala Thr Ile Val Ile Val Asp Ile Cys Ile Thr Gly Gly Leu
130 135 140
Leu Leu Leu Val Tyr Tyr Trp Ser Lys Asn Arg Lys Ala Lys Ala Lys
145 150 155 160
Pro Val Thr Arg Gly Ala Gly Ala Gly Gly Arg Gln Arg Gly Gln Asn
165 170 175
Lys Glu Arg Pro Pro Pro Val Pro Asn Pro Asp Tyr Glu Pro Ile Arg
180 185 190
Lys Gly Gln Arg Asp Leu Tyr Ser Gly Leu Asn Gln Arg Arg Ile
195 200 205
<210> 33
<211> 523
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 33
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys
20 25 30
Pro Gly Ala Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe
35 40 45
Thr Asp Tyr Glu Met His Trp Ile Arg Gln Pro Pro Gly Gln Gly Leu
50 55 60
Glu Trp Ile Gly Ala Ile Asp Pro Lys Thr Gly Asp Thr Ala Tyr Ser
65 70 75 80
Gln Lys Phe Lys Gly Arg Val Thr Leu Thr Ala Asp Lys Ser Thr Ser
85 90 95
Thr Ala Tyr Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Thr Arg Phe Tyr Ser Tyr Thr Tyr Trp Gly Gln Gly Thr
115 120 125
Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
130 135 140
Gly Gly Gly Gly Ser Asp Ile Val Met Thr Gln Ser Pro Leu Ser Leu
145 150 155 160
Pro Val Thr Pro Gly Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln
165 170 175
Ser Leu Val His Ser Asn Arg Asn Thr Tyr Leu His Trp Tyr Gln Gln
180 185 190
Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr Lys Val Ser Asn Arg
195 200 205
Phe Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp
210 215 220
Phe Thr Leu Lys Ile Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr
225 230 235 240
Tyr Cys Ser Gln Asn Thr His Val Pro Pro Thr Phe Gly Gln Gly Thr
245 250 255
Lys Leu Glu Ile Lys Gly Gly Gly Gly Ser Gln Ala Val Val Thr Gln
260 265 270
Glu Ser Ala Leu Thr Thr Ser Pro Gly Glu Thr Val Thr Leu Thr Cys
275 280 285
Arg Ser Ser Thr Gly Ala Val Thr Thr Ser Asn Tyr Ala Asn Trp Val
290 295 300
Gln Glu Lys Pro Asp His Leu Phe Thr Gly Leu Ile Gly Gly Thr Asn
305 310 315 320
Lys Arg Ala Pro Gly Val Pro Ala Arg Phe Ser Gly Ser Leu Ile Gly
325 330 335
Asp Lys Ala Ala Leu Thr Ile Thr Gly Ala Gln Thr Glu Asp Glu Ala
340 345 350
Ile Tyr Phe Cys Ala Leu Trp Tyr Ser Asn Leu Trp Val Phe Gly Gly
355 360 365
Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly Ser Gly Gly Gly Gly
370 375 380
Ser Gly Gly Gly Gly Ser Glu Val Lys Leu Leu Glu Ser Gly Gly Gly
385 390 395 400
Leu Val Gln Pro Lys Gly Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly
405 410 415
Phe Thr Phe Asn Thr Tyr Ala Met Asn Trp Val Arg Gln Ala Pro Gly
420 425 430
Lys Gly Leu Glu Trp Val Ala Arg Ile Arg Ser Lys Tyr Asn Asn Tyr
435 440 445
Ala Thr Tyr Tyr Ala Asp Ser Val Lys Asp Arg Phe Thr Ile Ser Arg
450 455 460
Asp Asp Ser Gln Ser Ile Leu Tyr Leu Gln Met Asn Asn Leu Lys Thr
465 470 475 480
Glu Asp Thr Ala Met Tyr Tyr Cys Val Arg His Gly Asn Phe Gly Asn
485 490 495
Ser Tyr Val Ser Trp Phe Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr
500 505 510
Val Ser Ala His His His His His His His His
515 520
<210> 34
<211> 753
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 34
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys
20 25 30
Pro Gly Ala Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe
35 40 45
Thr Asp Tyr Glu Met His Trp Ile Arg Gln Pro Pro Gly Gln Gly Leu
50 55 60
Glu Trp Ile Gly Ala Ile Asp Pro Lys Thr Gly Asp Thr Ala Tyr Ser
65 70 75 80
Gln Lys Phe Lys Gly Arg Val Thr Leu Thr Ala Asp Lys Ser Thr Ser
85 90 95
Thr Ala Tyr Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Thr Arg Phe Tyr Ser Tyr Thr Tyr Trp Gly Gln Gly Thr
115 120 125
Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
130 135 140
Gly Gly Gly Gly Ser Asp Ile Val Met Thr Gln Ser Pro Leu Ser Leu
145 150 155 160
Pro Val Thr Pro Gly Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln
165 170 175
Ser Leu Val His Ser Asn Arg Asn Thr Tyr Leu His Trp Tyr Gln Gln
180 185 190
Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr Lys Val Ser Asn Arg
195 200 205
Phe Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp
210 215 220
Phe Thr Leu Lys Ile Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr
225 230 235 240
Tyr Cys Ser Gln Asn Thr His Val Pro Pro Thr Phe Gly Gln Gly Thr
245 250 255
Lys Leu Glu Ile Lys Gly Gly Gly Gly Ser Gln Ala Val Val Thr Gln
260 265 270
Glu Ser Ala Leu Thr Thr Ser Pro Gly Glu Thr Val Thr Leu Thr Cys
275 280 285
Arg Ser Ser Thr Gly Ala Val Thr Thr Ser Asn Tyr Ala Asn Trp Val
290 295 300
Gln Glu Lys Pro Asp His Leu Phe Thr Gly Leu Ile Gly Gly Thr Asn
305 310 315 320
Lys Arg Ala Pro Gly Val Pro Ala Arg Phe Ser Gly Ser Leu Ile Gly
325 330 335
Asp Lys Ala Ala Leu Thr Ile Thr Gly Ala Gln Thr Glu Asp Glu Ala
340 345 350
Ile Tyr Phe Cys Ala Leu Trp Tyr Ser Asn Leu Trp Val Phe Gly Gly
355 360 365
Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly Ser Gly Gly Gly Gly
370 375 380
Ser Gly Gly Gly Gly Ser Glu Val Lys Leu Leu Glu Ser Gly Gly Gly
385 390 395 400
Leu Val Gln Pro Lys Gly Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly
405 410 415
Phe Thr Phe Asn Thr Tyr Ala Met Asn Trp Val Arg Gln Ala Pro Gly
420 425 430
Lys Gly Leu Glu Trp Val Ala Arg Ile Arg Ser Lys Tyr Asn Asn Tyr
435 440 445
Ala Thr Tyr Tyr Ala Asp Ser Val Lys Asp Arg Phe Thr Ile Ser Arg
450 455 460
Asp Asp Ser Gln Ser Ile Leu Tyr Leu Gln Met Asn Asn Leu Lys Thr
465 470 475 480
Glu Asp Thr Ala Met Tyr Tyr Cys Val Arg His Gly Asn Phe Gly Asn
485 490 495
Ser Tyr Val Ser Trp Phe Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr
500 505 510
Val Ser Ala Glu Pro Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro
515 520 525
Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro
530 535 540
Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr
545 550 555 560
Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn
565 570 575
Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg
580 585 590
Glu Glu Gln Tyr Ala Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val
595 600 605
Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser
610 615 620
Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys
625 630 635 640
Gly Gln Pro Arg Glu Pro Gln Val Cys Thr Leu Pro Pro Ser Arg Asp
645 650 655
Glu Leu Thr Lys Asn Gln Val Ser Leu Trp Cys Leu Val Lys Gly Phe
660 665 670
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
675 680 685
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
690 695 700
Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly
705 710 715 720
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
725 730 735
Thr Gln Lys Ser Leu Ser Leu Ser Pro His His His His His His His
740 745 750
His
<210> 35
<211> 257
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 35
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Glu Pro Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro
20 25 30
Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro
35 40 45
Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr
50 55 60
Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn
65 70 75 80
Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg
85 90 95
Glu Glu Gln Tyr Ala Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val
100 105 110
Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser
115 120 125
Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys
130 135 140
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Cys
145 150 155 160
Glu Leu Thr Lys Asn Gln Val Ser Leu Ser Cys Ala Val Lys Gly Phe
165 170 175
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
180 185 190
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
195 200 205
Phe Leu Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly
210 215 220
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
225 230 235 240
Thr Gln Lys Ser Leu Ser Leu Ser Pro Asp Tyr Lys Asp Asp Asp Asp
245 250 255
Lys
<210> 36
<211> 514
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 36
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys
20 25 30
Pro Gly Ala Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe
35 40 45
Thr Asp Tyr Glu Met His Trp Ile Arg Gln Pro Pro Gly Gln Gly Leu
50 55 60
Glu Trp Ile Gly Ala Ile Asp Pro Lys Thr Gly Asp Thr Ala Tyr Ser
65 70 75 80
Gln Lys Phe Lys Gly Arg Val Thr Leu Thr Ala Asp Lys Ser Thr Ser
85 90 95
Thr Ala Tyr Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Thr Arg Phe Tyr Ser Tyr Thr Tyr Trp Gly Gln Gly Thr
115 120 125
Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
130 135 140
Gly Gly Gly Gly Ser Asp Ile Val Met Thr Gln Ser Pro Leu Ser Leu
145 150 155 160
Pro Val Thr Pro Gly Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln
165 170 175
Ser Leu Val His Ser Asn Arg Asn Thr Tyr Leu His Trp Tyr Gln Gln
180 185 190
Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr Lys Val Ser Asn Arg
195 200 205
Phe Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp
210 215 220
Phe Thr Leu Lys Ile Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr
225 230 235 240
Tyr Cys Ser Gln Asn Thr His Val Pro Pro Thr Phe Gly Gln Gly Thr
245 250 255
Lys Leu Glu Ile Lys Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
260 265 270
Gly Gly Gly Ser Glu Pro Lys Ser Ser Asp Lys Thr His Thr Cys Pro
275 280 285
Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe
290 295 300
Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val
305 310 315 320
Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe
325 330 335
Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro
340 345 350
Arg Glu Glu Gln Tyr Ala Ser Thr Tyr Arg Val Val Ser Val Leu Thr
355 360 365
Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val
370 375 380
Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala
385 390 395 400
Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg
405 410 415
Cys Glu Leu Thr Lys Asn Gln Val Ser Leu Ser Cys Ala Val Lys Gly
420 425 430
Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro
435 440 445
Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser
450 455 460
Phe Phe Leu Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln
465 470 475 480
Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His
485 490 495
Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Asp Tyr Lys Asp Asp Asp
500 505 510
Asp Lys
<210> 37
<211> 629
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 37
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys
20 25 30
Pro Gly Ala Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe
35 40 45
Thr Asp Tyr Glu Met His Trp Ile Arg Gln Pro Pro Gly Gln Gly Leu
50 55 60
Glu Trp Ile Gly Ala Ile Asp Pro Lys Thr Gly Asp Thr Ala Tyr Ser
65 70 75 80
Gln Lys Phe Lys Gly Arg Val Thr Leu Thr Ala Asp Lys Ser Thr Ser
85 90 95
Thr Ala Tyr Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Thr Arg Phe Tyr Ser Tyr Thr Tyr Trp Gly Gln Gly Thr
115 120 125
Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
130 135 140
Gly Gly Gly Gly Ser Asp Ile Val Met Thr Gln Ser Pro Leu Ser Leu
145 150 155 160
Pro Val Thr Pro Gly Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln
165 170 175
Ser Leu Val His Ser Asn Arg Asn Thr Tyr Leu His Trp Tyr Gln Gln
180 185 190
Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr Lys Val Ser Asn Arg
195 200 205
Phe Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp
210 215 220
Phe Thr Leu Lys Ile Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr
225 230 235 240
Tyr Cys Ser Gln Asn Thr His Val Pro Pro Thr Phe Gly Gln Gly Thr
245 250 255
Lys Leu Glu Ile Lys Gly Gly Gly Gly Ser Glu Val Lys Leu Leu Glu
260 265 270
Ser Gly Gly Gly Leu Val Gln Pro Lys Gly Ser Leu Lys Leu Ser Cys
275 280 285
Ala Ala Ser Gly Phe Thr Phe Asn Thr Tyr Ala Met Asn Trp Val Arg
290 295 300
Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Arg Ile Arg Ser Lys
305 310 315 320
Tyr Asn Asn Tyr Ala Thr Tyr Tyr Ala Asp Ser Val Lys Asp Arg Phe
325 330 335
Thr Ile Ser Arg Asp Asp Ser Gln Ser Ile Leu Tyr Leu Gln Met Asn
340 345 350
Asn Leu Lys Thr Glu Asp Thr Ala Met Tyr Tyr Cys Val Arg His Gly
355 360 365
Asn Phe Gly Asn Ser Tyr Val Ser Trp Phe Ala Tyr Trp Gly Gln Gly
370 375 380
Thr Leu Val Thr Val Ser Ala Glu Pro Lys Ser Ser Asp Lys Thr His
385 390 395 400
Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val
405 410 415
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
420 425 430
Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu
435 440 445
Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
450 455 460
Thr Lys Pro Arg Glu Glu Gln Tyr Ala Ser Thr Tyr Arg Val Val Ser
465 470 475 480
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
485 490 495
Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile
500 505 510
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Cys Thr Leu Pro
515 520 525
Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Trp Cys Leu
530 535 540
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
545 550 555 560
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
565 570 575
Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg
580 585 590
Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
595 600 605
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro His His His
610 615 620
His His His His His
625
<210> 38
<211> 613
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 38
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys
20 25 30
Pro Gly Ala Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe
35 40 45
Thr Asp Tyr Glu Met His Trp Ile Arg Gln Pro Pro Gly Gln Gly Leu
50 55 60
Glu Trp Ile Gly Ala Ile Asp Pro Lys Thr Gly Asp Thr Ala Tyr Ser
65 70 75 80
Gln Lys Phe Lys Gly Arg Val Thr Leu Thr Ala Asp Lys Ser Thr Ser
85 90 95
Thr Ala Tyr Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Thr Arg Phe Tyr Ser Tyr Thr Tyr Trp Gly Gln Gly Thr
115 120 125
Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
130 135 140
Gly Gly Gly Gly Ser Asp Ile Val Met Thr Gln Ser Pro Leu Ser Leu
145 150 155 160
Pro Val Thr Pro Gly Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln
165 170 175
Ser Leu Val His Ser Asn Arg Asn Thr Tyr Leu His Trp Tyr Gln Gln
180 185 190
Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr Lys Val Ser Asn Arg
195 200 205
Phe Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp
210 215 220
Phe Thr Leu Lys Ile Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr
225 230 235 240
Tyr Cys Ser Gln Asn Thr His Val Pro Pro Thr Phe Gly Gln Gly Thr
245 250 255
Lys Leu Glu Ile Lys Gly Gly Gly Gly Ser Gln Ala Val Val Thr Gln
260 265 270
Glu Ser Ala Leu Thr Thr Ser Pro Gly Glu Thr Val Thr Leu Thr Cys
275 280 285
Arg Ser Ser Thr Gly Ala Val Thr Thr Ser Asn Tyr Ala Asn Trp Val
290 295 300
Gln Glu Lys Pro Asp His Leu Phe Thr Gly Leu Ile Gly Gly Thr Asn
305 310 315 320
Lys Arg Ala Pro Gly Val Pro Ala Arg Phe Ser Gly Ser Leu Ile Gly
325 330 335
Asp Lys Ala Ala Leu Thr Ile Thr Gly Ala Gln Thr Glu Asp Glu Ala
340 345 350
Ile Tyr Phe Cys Ala Leu Trp Tyr Ser Asn Leu Trp Val Phe Gly Gly
355 360 365
Gly Thr Lys Leu Thr Val Leu Glu Pro Lys Ser Ser Asp Lys Thr His
370 375 380
Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val
385 390 395 400
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
405 410 415
Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu
420 425 430
Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
435 440 445
Thr Lys Pro Arg Glu Glu Gln Tyr Ala Ser Thr Tyr Arg Val Val Ser
450 455 460
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
465 470 475 480
Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile
485 490 495
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
500 505 510
Pro Ser Arg Cys Glu Leu Thr Lys Asn Gln Val Ser Leu Ser Cys Ala
515 520 525
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
530 535 540
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
545 550 555 560
Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val Asp Lys Ser Arg
565 570 575
Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
580 585 590
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Asp Tyr Lys
595 600 605
Asp Asp Asp Asp Lys
610
<210> 39
<211> 470
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 39
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys
20 25 30
Pro Gly Ala Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe
35 40 45
Thr Asp Tyr Glu Met His Trp Ile Arg Gln Pro Pro Gly Gln Gly Leu
50 55 60
Glu Trp Ile Gly Ala Ile Asp Pro Lys Thr Gly Asp Thr Ala Tyr Ser
65 70 75 80
Gln Lys Phe Lys Gly Arg Val Thr Leu Thr Ala Asp Lys Ser Thr Ser
85 90 95
Thr Ala Tyr Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Thr Arg Phe Tyr Ser Tyr Thr Tyr Trp Gly Gln Gly Thr
115 120 125
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
130 135 140
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
145 150 155 160
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
165 170 175
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
180 185 190
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
195 200 205
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
210 215 220
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
225 230 235 240
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser
245 250 255
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
260 265 270
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
275 280 285
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
290 295 300
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Ala Ser Thr Tyr Arg Val Val
305 310 315 320
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
325 330 335
Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr
340 345 350
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
355 360 365
Pro Pro Ser Arg Cys Glu Leu Thr Lys Asn Gln Val Ser Leu Ser Cys
370 375 380
Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
385 390 395 400
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
405 410 415
Ser Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val Asp Lys Ser
420 425 430
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
435 440 445
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Asp Tyr
450 455 460
Lys Asp Asp Asp Asp Lys
465 470
<210> 40
<211> 238
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 40
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Asp Ile Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val
20 25 30
Thr Pro Gly Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu
35 40 45
Val His Ser Asn Arg Asn Thr Tyr Leu His Trp Tyr Gln Gln Lys Pro
50 55 60
Gly Gln Ala Pro Arg Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser
65 70 75 80
Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
85 90 95
Leu Lys Ile Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys
100 105 110
Ser Gln Asn Thr His Val Pro Pro Thr Phe Gly Gln Gly Thr Lys Leu
115 120 125
Glu Ile Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro
130 135 140
Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu
145 150 155 160
Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn
165 170 175
Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser
180 185 190
Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala
195 200 205
Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly
210 215 220
Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230 235
<210> 41
<211> 470
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 41
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys
20 25 30
Pro Gly Ala Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe
35 40 45
Thr Asp Tyr Glu Met His Trp Ile Arg Gln Pro Pro Gly Gln Gly Leu
50 55 60
Glu Trp Ile Gly Ala Ile Asp Pro Lys Thr Gly Asp Thr Ala Tyr Ser
65 70 75 80
Gln Lys Phe Lys Gly Arg Val Thr Leu Thr Ala Asp Lys Ser Thr Ser
85 90 95
Thr Ala Tyr Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Thr Arg Phe Tyr Ser Tyr Thr Tyr Trp Gly Gln Gly Thr
115 120 125
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
130 135 140
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
145 150 155 160
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
165 170 175
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
180 185 190
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
195 200 205
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
210 215 220
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
225 230 235 240
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser
245 250 255
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
260 265 270
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
275 280 285
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
290 295 300
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Ala Ser Thr Tyr Arg Val Val
305 310 315 320
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
325 330 335
Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr
340 345 350
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Cys Thr Leu
355 360 365
Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Trp Cys
370 375 380
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
385 390 395 400
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
405 410 415
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser
420 425 430
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
435 440 445
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro His His
450 455 460
His His His His His His
465 470
<210> 42
<211> 734
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 42
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Ala Val Val Thr Gln Glu Ser Ala Leu Thr Thr Ser
20 25 30
Pro Gly Glu Thr Val Thr Leu Thr Cys Arg Ser Ser Thr Gly Ala Val
35 40 45
Thr Thr Ser Asn Tyr Ala Asn Trp Val Gln Glu Lys Pro Asp His Leu
50 55 60
Phe Thr Gly Leu Ile Gly Gly Thr Asn Lys Arg Ala Pro Gly Val Pro
65 70 75 80
Ala Arg Phe Ser Gly Ser Leu Ile Gly Asp Lys Ala Ala Leu Thr Ile
85 90 95
Thr Gly Ala Gln Thr Glu Asp Glu Ala Ile Tyr Phe Cys Ala Leu Trp
100 105 110
Tyr Ser Asn Leu Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
115 120 125
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu
130 135 140
Val Lys Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Lys Gly Ser
145 150 155 160
Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asn Thr Tyr Ala
165 170 175
Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala
180 185 190
Arg Ile Arg Ser Lys Tyr Asn Asn Tyr Ala Thr Tyr Tyr Ala Asp Ser
195 200 205
Val Lys Asp Arg Phe Thr Ile Ser Arg Asp Asp Ser Gln Ser Ile Leu
210 215 220
Tyr Leu Gln Met Asn Asn Leu Lys Thr Glu Asp Thr Ala Met Tyr Tyr
225 230 235 240
Cys Val Arg His Gly Asn Phe Gly Asn Ser Tyr Val Ser Trp Phe Ala
245 250 255
Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ala Gly Gly Gly Gly
260 265 270
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln Leu Val
275 280 285
Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala Ser Val Lys Val Ser
290 295 300
Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr Glu Met His Trp Ile
305 310 315 320
Arg Gln Pro Pro Gly Gln Gly Leu Glu Trp Ile Gly Ala Ile Asp Pro
325 330 335
Lys Thr Gly Asp Thr Ala Tyr Ser Gln Lys Phe Lys Gly Arg Val Thr
340 345 350
Leu Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr Met Glu Leu Ser Ser
355 360 365
Leu Thr Ser Glu Asp Thr Ala Val Tyr Tyr Cys Thr Arg Phe Tyr Ser
370 375 380
Tyr Thr Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser
385 390 395 400
Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr
405 410 415
Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro
420 425 430
Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val
435 440 445
His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser
450 455 460
Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile
465 470 475 480
Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val
485 490 495
Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala
500 505 510
Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro
515 520 525
Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val
530 535 540
Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val
545 550 555 560
Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln
565 570 575
Tyr Ala Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln
580 585 590
Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala
595 600 605
Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro
610 615 620
Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Cys Glu Leu Thr
625 630 635 640
Lys Asn Gln Val Ser Leu Ser Cys Ala Val Lys Gly Phe Tyr Pro Ser
645 650 655
Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr
660 665 670
Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Val
675 680 685
Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe
690 695 700
Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys
705 710 715 720
Ser Leu Ser Leu Ser Pro Asp Tyr Lys Asp Asp Asp Asp Lys
725 730
<210> 43
<211> 462
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 43
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys
20 25 30
Pro Gly Ala Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe
35 40 45
Thr Asp Tyr Glu Met His Trp Ile Arg Gln Pro Pro Gly Gln Gly Leu
50 55 60
Glu Trp Ile Gly Ala Ile Asp Pro Lys Thr Gly Asp Thr Ala Tyr Ser
65 70 75 80
Gln Lys Phe Lys Gly Arg Val Thr Leu Thr Ala Asp Lys Ser Thr Ser
85 90 95
Thr Ala Tyr Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Thr Arg Phe Tyr Ser Tyr Thr Tyr Trp Gly Gln Gly Thr
115 120 125
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
130 135 140
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
145 150 155 160
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
165 170 175
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
180 185 190
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
195 200 205
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
210 215 220
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
225 230 235 240
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser
245 250 255
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
260 265 270
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
275 280 285
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
290 295 300
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Ala Ser Thr Tyr Arg Val Val
305 310 315 320
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
325 330 335
Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr
340 345 350
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
355 360 365
Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys
370 375 380
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
385 390 395 400
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
405 410 415
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser
420 425 430
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
435 440 445
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
450 455 460
<210> 44
<211> 246
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 44
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Asp Ile Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val
20 25 30
Thr Pro Gly Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu
35 40 45
Val His Ser Asn Arg Asn Thr Tyr Leu His Trp Tyr Gln Gln Lys Pro
50 55 60
Gly Gln Ala Pro Arg Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser
65 70 75 80
Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
85 90 95
Leu Lys Ile Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys
100 105 110
Ser Gln Asn Thr His Val Pro Pro Thr Phe Gly Gln Gly Thr Lys Leu
115 120 125
Glu Ile Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro
130 135 140
Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu
145 150 155 160
Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn
165 170 175
Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser
180 185 190
Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala
195 200 205
Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly
210 215 220
Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys His His
225 230 235 240
His His His His His His
245
<210> 45
<211> 505
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 45
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Asp Ile Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val
20 25 30
Thr Pro Gly Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu
35 40 45
Val His Ser Asn Arg Asn Thr Tyr Leu His Trp Tyr Gln Gln Lys Pro
50 55 60
Gly Gln Ala Pro Arg Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser
65 70 75 80
Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
85 90 95
Leu Lys Ile Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys
100 105 110
Ser Gln Asn Thr His Val Pro Pro Thr Phe Gly Gln Gly Thr Lys Leu
115 120 125
Glu Ile Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro
130 135 140
Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu
145 150 155 160
Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn
165 170 175
Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser
180 185 190
Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala
195 200 205
Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly
210 215 220
Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys Gly Gly
225 230 235 240
Gly Gly Ser Gly Gly Gly Gly Ser Gln Ala Val Val Thr Gln Glu Ser
245 250 255
Ala Leu Thr Thr Ser Pro Gly Glu Thr Val Thr Leu Thr Cys Arg Ser
260 265 270
Ser Thr Gly Ala Val Thr Thr Ser Asn Tyr Ala Asn Trp Val Gln Glu
275 280 285
Lys Pro Asp His Leu Phe Thr Gly Leu Ile Gly Gly Thr Asn Lys Arg
290 295 300
Ala Pro Gly Val Pro Ala Arg Phe Ser Gly Ser Leu Ile Gly Asp Lys
305 310 315 320
Ala Ala Leu Thr Ile Thr Gly Ala Gln Thr Glu Asp Glu Ala Ile Tyr
325 330 335
Phe Cys Ala Leu Trp Tyr Ser Asn Leu Trp Val Phe Gly Gly Gly Thr
340 345 350
Lys Leu Thr Val Leu Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
355 360 365
Gly Gly Gly Ser Glu Val Lys Leu Leu Glu Ser Gly Gly Gly Leu Val
370 375 380
Gln Pro Lys Gly Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Thr
385 390 395 400
Phe Asn Thr Tyr Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly
405 410 415
Leu Glu Trp Val Ala Arg Ile Arg Ser Lys Tyr Asn Asn Tyr Ala Thr
420 425 430
Tyr Tyr Ala Asp Ser Val Lys Asp Arg Phe Thr Ile Ser Arg Asp Asp
435 440 445
Ser Gln Ser Ile Leu Tyr Leu Gln Met Asn Asn Leu Lys Thr Glu Asp
450 455 460
Thr Ala Met Tyr Tyr Cys Val Arg His Gly Asn Phe Gly Asn Ser Tyr
465 470 475 480
Val Ser Trp Phe Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser
485 490 495
Ala Asp Tyr Lys Asp Asp Asp Asp Lys
500 505
<210> 46
<211> 729
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 46
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys
20 25 30
Pro Gly Ala Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe
35 40 45
Thr Asp Tyr Glu Met His Trp Ile Arg Gln Pro Pro Gly Gln Gly Leu
50 55 60
Glu Trp Ile Gly Ala Ile Asp Pro Lys Thr Gly Asp Thr Ala Tyr Ser
65 70 75 80
Gln Lys Phe Lys Gly Arg Val Thr Leu Thr Ala Asp Lys Ser Thr Ser
85 90 95
Thr Ala Tyr Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Thr Arg Phe Tyr Ser Tyr Thr Tyr Trp Gly Gln Gly Thr
115 120 125
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
130 135 140
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
145 150 155 160
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
165 170 175
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
180 185 190
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
195 200 205
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
210 215 220
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
225 230 235 240
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser
245 250 255
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
260 265 270
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
275 280 285
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
290 295 300
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Ala Ser Thr Tyr Arg Val Val
305 310 315 320
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
325 330 335
Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr
340 345 350
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
355 360 365
Pro Pro Ser Arg Cys Glu Leu Thr Lys Asn Gln Val Ser Leu Ser Cys
370 375 380
Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
385 390 395 400
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
405 410 415
Ser Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val Asp Lys Ser
420 425 430
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
435 440 445
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Gly
450 455 460
Gly Gly Ser Gly Gly Gly Gly Ser Gln Ala Val Val Thr Gln Glu Ser
465 470 475 480
Ala Leu Thr Thr Ser Pro Gly Glu Thr Val Thr Leu Thr Cys Arg Ser
485 490 495
Ser Thr Gly Ala Val Thr Thr Ser Asn Tyr Ala Asn Trp Val Gln Glu
500 505 510
Lys Pro Asp His Leu Phe Thr Gly Leu Ile Gly Gly Thr Asn Lys Arg
515 520 525
Ala Pro Gly Val Pro Ala Arg Phe Ser Gly Ser Leu Ile Gly Asp Lys
530 535 540
Ala Ala Leu Thr Ile Thr Gly Ala Gln Thr Glu Asp Glu Ala Ile Tyr
545 550 555 560
Phe Cys Ala Leu Trp Tyr Ser Asn Leu Trp Val Phe Gly Gly Gly Thr
565 570 575
Lys Leu Thr Val Leu Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
580 585 590
Gly Gly Gly Ser Glu Val Lys Leu Leu Glu Ser Gly Gly Gly Leu Val
595 600 605
Gln Pro Lys Gly Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Thr
610 615 620
Phe Asn Thr Tyr Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly
625 630 635 640
Leu Glu Trp Val Ala Arg Ile Arg Ser Lys Tyr Asn Asn Tyr Ala Thr
645 650 655
Tyr Tyr Ala Asp Ser Val Lys Asp Arg Phe Thr Ile Ser Arg Asp Asp
660 665 670
Ser Gln Ser Ile Leu Tyr Leu Gln Met Asn Asn Leu Lys Thr Glu Asp
675 680 685
Thr Ala Met Tyr Tyr Cys Val Arg His Gly Asn Phe Gly Asn Ser Tyr
690 695 700
Val Ser Trp Phe Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser
705 710 715 720
Ala Asp Tyr Lys Asp Asp Asp Asp Lys
725
<210> 47
<211> 729
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 47
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys
20 25 30
Pro Gly Ala Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe
35 40 45
Thr Asp Tyr Glu Met His Trp Ile Arg Gln Pro Pro Gly Gln Gly Leu
50 55 60
Glu Trp Ile Gly Ala Ile Asp Pro Lys Thr Gly Asp Thr Ala Tyr Ser
65 70 75 80
Gln Lys Phe Lys Gly Arg Val Thr Leu Thr Ala Asp Lys Ser Thr Ser
85 90 95
Thr Ala Tyr Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Thr Arg Phe Tyr Ser Tyr Thr Tyr Trp Gly Gln Gly Thr
115 120 125
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
130 135 140
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
145 150 155 160
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
165 170 175
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
180 185 190
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
195 200 205
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
210 215 220
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
225 230 235 240
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser
245 250 255
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
260 265 270
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
275 280 285
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
290 295 300
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val
305 310 315 320
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
325 330 335
Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr
340 345 350
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
355 360 365
Pro Pro Ser Arg Cys Glu Leu Thr Lys Asn Gln Val Ser Leu Ser Cys
370 375 380
Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
385 390 395 400
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
405 410 415
Ser Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val Asp Lys Ser
420 425 430
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
435 440 445
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Gly
450 455 460
Gly Gly Ser Gly Gly Gly Gly Ser Gln Ala Val Val Thr Gln Glu Ser
465 470 475 480
Ala Leu Thr Thr Ser Pro Gly Glu Thr Val Thr Leu Thr Cys Arg Ser
485 490 495
Ser Thr Gly Ala Val Thr Thr Ser Asn Tyr Ala Asn Trp Val Gln Glu
500 505 510
Lys Pro Asp His Leu Phe Thr Gly Leu Ile Gly Gly Thr Asn Lys Arg
515 520 525
Ala Pro Gly Val Pro Ala Arg Phe Ser Gly Ser Leu Ile Gly Asp Lys
530 535 540
Ala Ala Leu Thr Ile Thr Gly Ala Gln Thr Glu Asp Glu Ala Ile Tyr
545 550 555 560
Phe Cys Ala Leu Trp Tyr Ser Asn Leu Trp Val Phe Gly Gly Gly Thr
565 570 575
Lys Leu Thr Val Leu Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
580 585 590
Gly Gly Gly Ser Glu Val Lys Leu Leu Glu Ser Gly Gly Gly Leu Val
595 600 605
Gln Pro Lys Gly Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Thr
610 615 620
Phe Asn Thr Tyr Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly
625 630 635 640
Leu Glu Trp Val Ala Arg Ile Arg Ser Lys Tyr Asn Asn Tyr Ala Thr
645 650 655
Tyr Tyr Ala Asp Ser Val Lys Asp Arg Phe Thr Ile Ser Arg Asp Asp
660 665 670
Ser Gln Ser Ile Leu Tyr Leu Gln Met Asn Asn Leu Lys Thr Glu Asp
675 680 685
Thr Ala Met Tyr Tyr Cys Val Arg His Gly Asn Phe Gly Asn Ser Tyr
690 695 700
Val Ser Trp Phe Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser
705 710 715 720
Ala Asp Tyr Lys Asp Asp Asp Asp Lys
725
<210> 48
<211> 470
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 48
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys
20 25 30
Pro Gly Ala Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe
35 40 45
Thr Asp Tyr Glu Met His Trp Ile Arg Gln Pro Pro Gly Gln Gly Leu
50 55 60
Glu Trp Ile Gly Ala Ile Asp Pro Lys Thr Gly Asp Thr Ala Tyr Ser
65 70 75 80
Gln Lys Phe Lys Gly Arg Val Thr Leu Thr Ala Asp Lys Ser Thr Ser
85 90 95
Thr Ala Tyr Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Thr Arg Phe Tyr Ser Tyr Thr Tyr Trp Gly Gln Gly Thr
115 120 125
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
130 135 140
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
145 150 155 160
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
165 170 175
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
180 185 190
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
195 200 205
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
210 215 220
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
225 230 235 240
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser
245 250 255
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
260 265 270
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
275 280 285
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
290 295 300
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val
305 310 315 320
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
325 330 335
Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr
340 345 350
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Cys Thr Leu
355 360 365
Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Trp Cys
370 375 380
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
385 390 395 400
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
405 410 415
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser
420 425 430
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
435 440 445
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro His His
450 455 460
His His His His His His
465 470
<210> 49
<211> 600
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 49
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys
20 25 30
Pro Gly Ala Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe
35 40 45
Thr Asp Tyr Glu Met His Trp Ile Arg Gln Pro Pro Gly Gln Gly Leu
50 55 60
Glu Trp Ile Gly Ala Ile Asp Pro Lys Thr Gly Asp Thr Ala Tyr Ser
65 70 75 80
Gln Lys Phe Lys Gly Arg Val Thr Leu Thr Ala Asp Lys Ser Thr Ser
85 90 95
Thr Ala Tyr Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Thr Arg Phe Tyr Ser Tyr Thr Tyr Trp Gly Gln Gly Thr
115 120 125
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
130 135 140
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
145 150 155 160
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
165 170 175
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
180 185 190
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
195 200 205
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
210 215 220
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
225 230 235 240
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser
245 250 255
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
260 265 270
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
275 280 285
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
290 295 300
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Ala Ser Thr Tyr Arg Val Val
305 310 315 320
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
325 330 335
Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr
340 345 350
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
355 360 365
Pro Pro Ser Arg Cys Glu Leu Thr Lys Asn Gln Val Ser Leu Ser Cys
370 375 380
Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
385 390 395 400
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
405 410 415
Ser Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val Asp Lys Ser
420 425 430
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
435 440 445
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Gly
450 455 460
Gly Gly Ser Glu Val Lys Leu Leu Glu Ser Gly Gly Gly Leu Val Gln
465 470 475 480
Pro Lys Gly Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
485 490 495
Asn Thr Tyr Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
500 505 510
Glu Trp Val Ala Arg Ile Arg Ser Lys Tyr Asn Asn Tyr Ala Thr Tyr
515 520 525
Tyr Ala Asp Ser Val Lys Asp Arg Phe Thr Ile Ser Arg Asp Asp Ser
530 535 540
Gln Ser Ile Leu Tyr Leu Gln Met Asn Asn Leu Lys Thr Glu Asp Thr
545 550 555 560
Ala Met Tyr Tyr Cys Val Arg His Gly Asn Phe Gly Asn Ser Tyr Val
565 570 575
Ser Trp Phe Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ala
580 585 590
Asp Tyr Lys Asp Asp Asp Asp Lys
595 600
<210> 50
<211> 584
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 50
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys
20 25 30
Pro Gly Ala Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe
35 40 45
Thr Asp Tyr Glu Met His Trp Ile Arg Gln Pro Pro Gly Gln Gly Leu
50 55 60
Glu Trp Ile Gly Ala Ile Asp Pro Lys Thr Gly Asp Thr Ala Tyr Ser
65 70 75 80
Gln Lys Phe Lys Gly Arg Val Thr Leu Thr Ala Asp Lys Ser Thr Ser
85 90 95
Thr Ala Tyr Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Thr Arg Phe Tyr Ser Tyr Thr Tyr Trp Gly Gln Gly Thr
115 120 125
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
130 135 140
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
145 150 155 160
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
165 170 175
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
180 185 190
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
195 200 205
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
210 215 220
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
225 230 235 240
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser
245 250 255
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
260 265 270
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
275 280 285
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
290 295 300
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Ala Ser Thr Tyr Arg Val Val
305 310 315 320
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
325 330 335
Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr
340 345 350
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Cys Thr Leu
355 360 365
Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Trp Cys
370 375 380
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
385 390 395 400
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
405 410 415
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser
420 425 430
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
435 440 445
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Gly
450 455 460
Gly Gly Ser Gln Ala Val Val Thr Gln Glu Ser Ala Leu Thr Thr Ser
465 470 475 480
Pro Gly Glu Thr Val Thr Leu Thr Cys Arg Ser Ser Thr Gly Ala Val
485 490 495
Thr Thr Ser Asn Tyr Ala Asn Trp Val Gln Glu Lys Pro Asp His Leu
500 505 510
Phe Thr Gly Leu Ile Gly Gly Thr Asn Lys Arg Ala Pro Gly Val Pro
515 520 525
Ala Arg Phe Ser Gly Ser Leu Ile Gly Asp Lys Ala Ala Leu Thr Ile
530 535 540
Thr Gly Ala Gln Thr Glu Asp Glu Ala Ile Tyr Phe Cys Ala Leu Trp
545 550 555 560
Tyr Ser Asn Leu Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
565 570 575
His His His His His His His His
580
<210> 51
<211> 605
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 51
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys
20 25 30
Pro Gly Ala Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe
35 40 45
Thr Asp Tyr Glu Met His Trp Ile Arg Gln Pro Pro Gly Gln Gly Leu
50 55 60
Glu Trp Ile Gly Ala Ile Asp Pro Lys Thr Gly Asp Thr Ala Tyr Ser
65 70 75 80
Gln Lys Phe Lys Gly Arg Val Thr Leu Thr Ala Asp Lys Ser Thr Ser
85 90 95
Thr Ala Tyr Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Thr Arg Phe Tyr Ser Tyr Thr Tyr Trp Gly Gln Gly Thr
115 120 125
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
130 135 140
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
145 150 155 160
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
165 170 175
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
180 185 190
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
195 200 205
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
210 215 220
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
225 230 235 240
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser
245 250 255
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
260 265 270
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
275 280 285
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
290 295 300
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Ala Ser Thr Tyr Arg Val Val
305 310 315 320
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
325 330 335
Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr
340 345 350
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
355 360 365
Pro Pro Ser Arg Cys Glu Leu Thr Lys Asn Gln Val Ser Leu Ser Cys
370 375 380
Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
385 390 395 400
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
405 410 415
Ser Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val Asp Lys Ser
420 425 430
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
435 440 445
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Gly
450 455 460
Gly Gly Ser Gly Gly Gly Gly Ser Glu Val Lys Leu Leu Glu Ser Gly
465 470 475 480
Gly Gly Leu Val Gln Pro Lys Gly Ser Leu Lys Leu Ser Cys Ala Ala
485 490 495
Ser Gly Phe Thr Phe Asn Thr Tyr Ala Met Asn Trp Val Arg Gln Ala
500 505 510
Pro Gly Lys Gly Leu Glu Trp Val Ala Arg Ile Arg Ser Lys Tyr Asn
515 520 525
Asn Tyr Ala Thr Tyr Tyr Ala Asp Ser Val Lys Asp Arg Phe Thr Ile
530 535 540
Ser Arg Asp Asp Ser Gln Ser Ile Leu Tyr Leu Gln Met Asn Asn Leu
545 550 555 560
Lys Thr Glu Asp Thr Ala Met Tyr Tyr Cys Val Arg His Gly Asn Phe
565 570 575
Gly Asn Ser Tyr Val Ser Trp Phe Ala Tyr Trp Gly Gln Gly Thr Leu
580 585 590
Val Thr Val Ser Ala Asp Tyr Lys Asp Asp Asp Asp Lys
595 600 605
<210> 52
<211> 589
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 52
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys
20 25 30
Pro Gly Ala Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe
35 40 45
Thr Asp Tyr Glu Met His Trp Ile Arg Gln Pro Pro Gly Gln Gly Leu
50 55 60
Glu Trp Ile Gly Ala Ile Asp Pro Lys Thr Gly Asp Thr Ala Tyr Ser
65 70 75 80
Gln Lys Phe Lys Gly Arg Val Thr Leu Thr Ala Asp Lys Ser Thr Ser
85 90 95
Thr Ala Tyr Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Thr Arg Phe Tyr Ser Tyr Thr Tyr Trp Gly Gln Gly Thr
115 120 125
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
130 135 140
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
145 150 155 160
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
165 170 175
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
180 185 190
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
195 200 205
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
210 215 220
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
225 230 235 240
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser
245 250 255
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
260 265 270
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
275 280 285
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
290 295 300
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Ala Ser Thr Tyr Arg Val Val
305 310 315 320
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
325 330 335
Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr
340 345 350
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Cys Thr Leu
355 360 365
Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Trp Cys
370 375 380
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
385 390 395 400
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
405 410 415
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser
420 425 430
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
435 440 445
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Gly
450 455 460
Gly Gly Ser Gly Gly Gly Gly Ser Gln Ala Val Val Thr Gln Glu Ser
465 470 475 480
Ala Leu Thr Thr Ser Pro Gly Glu Thr Val Thr Leu Thr Cys Arg Ser
485 490 495
Ser Thr Gly Ala Val Thr Thr Ser Asn Tyr Ala Asn Trp Val Gln Glu
500 505 510
Lys Pro Asp His Leu Phe Thr Gly Leu Ile Gly Gly Thr Asn Lys Arg
515 520 525
Ala Pro Gly Val Pro Ala Arg Phe Ser Gly Ser Leu Ile Gly Asp Lys
530 535 540
Ala Ala Leu Thr Ile Thr Gly Ala Gln Thr Glu Asp Glu Ala Ile Tyr
545 550 555 560
Phe Cys Ala Leu Trp Tyr Ser Asn Leu Trp Val Phe Gly Gly Gly Thr
565 570 575
Lys Leu Thr Val Leu His His His His His His His His
580 585
<210> 53
<211> 610
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 53
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys
20 25 30
Pro Gly Ala Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe
35 40 45
Thr Asp Tyr Glu Met His Trp Ile Arg Gln Pro Pro Gly Gln Gly Leu
50 55 60
Glu Trp Ile Gly Ala Ile Asp Pro Lys Thr Gly Asp Thr Ala Tyr Ser
65 70 75 80
Gln Lys Phe Lys Gly Arg Val Thr Leu Thr Ala Asp Lys Ser Thr Ser
85 90 95
Thr Ala Tyr Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Thr Arg Phe Tyr Ser Tyr Thr Tyr Trp Gly Gln Gly Thr
115 120 125
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
130 135 140
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
145 150 155 160
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
165 170 175
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
180 185 190
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
195 200 205
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
210 215 220
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
225 230 235 240
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser
245 250 255
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
260 265 270
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
275 280 285
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
290 295 300
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Ala Ser Thr Tyr Arg Val Val
305 310 315 320
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
325 330 335
Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr
340 345 350
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
355 360 365
Pro Pro Ser Arg Cys Glu Leu Thr Lys Asn Gln Val Ser Leu Ser Cys
370 375 380
Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
385 390 395 400
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
405 410 415
Ser Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val Asp Lys Ser
420 425 430
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
435 440 445
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Gly
450 455 460
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Val Lys
465 470 475 480
Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Lys Gly Ser Leu Lys
485 490 495
Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asn Thr Tyr Ala Met Asn
500 505 510
Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Arg Ile
515 520 525
Arg Ser Lys Tyr Asn Asn Tyr Ala Thr Tyr Tyr Ala Asp Ser Val Lys
530 535 540
Asp Arg Phe Thr Ile Ser Arg Asp Asp Ser Gln Ser Ile Leu Tyr Leu
545 550 555 560
Gln Met Asn Asn Leu Lys Thr Glu Asp Thr Ala Met Tyr Tyr Cys Val
565 570 575
Arg His Gly Asn Phe Gly Asn Ser Tyr Val Ser Trp Phe Ala Tyr Trp
580 585 590
Gly Gln Gly Thr Leu Val Thr Val Ser Ala Asp Tyr Lys Asp Asp Asp
595 600 605
Asp Lys
610
<210> 54
<211> 594
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 54
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys
20 25 30
Pro Gly Ala Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe
35 40 45
Thr Asp Tyr Glu Met His Trp Ile Arg Gln Pro Pro Gly Gln Gly Leu
50 55 60
Glu Trp Ile Gly Ala Ile Asp Pro Lys Thr Gly Asp Thr Ala Tyr Ser
65 70 75 80
Gln Lys Phe Lys Gly Arg Val Thr Leu Thr Ala Asp Lys Ser Thr Ser
85 90 95
Thr Ala Tyr Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Thr Arg Phe Tyr Ser Tyr Thr Tyr Trp Gly Gln Gly Thr
115 120 125
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
130 135 140
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
145 150 155 160
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
165 170 175
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
180 185 190
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
195 200 205
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
210 215 220
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
225 230 235 240
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser
245 250 255
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
260 265 270
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
275 280 285
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
290 295 300
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Ala Ser Thr Tyr Arg Val Val
305 310 315 320
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
325 330 335
Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr
340 345 350
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Cys Thr Leu
355 360 365
Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Trp Cys
370 375 380
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
385 390 395 400
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
405 410 415
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser
420 425 430
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
435 440 445
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Gly
450 455 460
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ala Val
465 470 475 480
Val Thr Gln Glu Ser Ala Leu Thr Thr Ser Pro Gly Glu Thr Val Thr
485 490 495
Leu Thr Cys Arg Ser Ser Thr Gly Ala Val Thr Thr Ser Asn Tyr Ala
500 505 510
Asn Trp Val Gln Glu Lys Pro Asp His Leu Phe Thr Gly Leu Ile Gly
515 520 525
Gly Thr Asn Lys Arg Ala Pro Gly Val Pro Ala Arg Phe Ser Gly Ser
530 535 540
Leu Ile Gly Asp Lys Ala Ala Leu Thr Ile Thr Gly Ala Gln Thr Glu
545 550 555 560
Asp Glu Ala Ile Tyr Phe Cys Ala Leu Trp Tyr Ser Asn Leu Trp Val
565 570 575
Phe Gly Gly Gly Thr Lys Leu Thr Val Leu His His His His His His
580 585 590
His His
<210> 55
<211> 615
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 55
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys
20 25 30
Pro Gly Ala Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe
35 40 45
Thr Asp Tyr Glu Met His Trp Ile Arg Gln Pro Pro Gly Gln Gly Leu
50 55 60
Glu Trp Ile Gly Ala Ile Asp Pro Lys Thr Gly Asp Thr Ala Tyr Ser
65 70 75 80
Gln Lys Phe Lys Gly Arg Val Thr Leu Thr Ala Asp Lys Ser Thr Ser
85 90 95
Thr Ala Tyr Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Thr Arg Phe Tyr Ser Tyr Thr Tyr Trp Gly Gln Gly Thr
115 120 125
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
130 135 140
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
145 150 155 160
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
165 170 175
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
180 185 190
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
195 200 205
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
210 215 220
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
225 230 235 240
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser
245 250 255
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
260 265 270
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
275 280 285
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
290 295 300
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Ala Ser Thr Tyr Arg Val Val
305 310 315 320
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
325 330 335
Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr
340 345 350
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
355 360 365
Pro Pro Ser Arg Cys Glu Leu Thr Lys Asn Gln Val Ser Leu Ser Cys
370 375 380
Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
385 390 395 400
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
405 410 415
Ser Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val Asp Lys Ser
420 425 430
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
435 440 445
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Gly
450 455 460
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
465 470 475 480
Gly Ser Glu Val Lys Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro
485 490 495
Lys Gly Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asn
500 505 510
Thr Tyr Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu
515 520 525
Trp Val Ala Arg Ile Arg Ser Lys Tyr Asn Asn Tyr Ala Thr Tyr Tyr
530 535 540
Ala Asp Ser Val Lys Asp Arg Phe Thr Ile Ser Arg Asp Asp Ser Gln
545 550 555 560
Ser Ile Leu Tyr Leu Gln Met Asn Asn Leu Lys Thr Glu Asp Thr Ala
565 570 575
Met Tyr Tyr Cys Val Arg His Gly Asn Phe Gly Asn Ser Tyr Val Ser
580 585 590
Trp Phe Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ala Asp
595 600 605
Tyr Lys Asp Asp Asp Asp Lys
610 615
<210> 56
<211> 599
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 56
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys
20 25 30
Pro Gly Ala Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe
35 40 45
Thr Asp Tyr Glu Met His Trp Ile Arg Gln Pro Pro Gly Gln Gly Leu
50 55 60
Glu Trp Ile Gly Ala Ile Asp Pro Lys Thr Gly Asp Thr Ala Tyr Ser
65 70 75 80
Gln Lys Phe Lys Gly Arg Val Thr Leu Thr Ala Asp Lys Ser Thr Ser
85 90 95
Thr Ala Tyr Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Thr Arg Phe Tyr Ser Tyr Thr Tyr Trp Gly Gln Gly Thr
115 120 125
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
130 135 140
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
145 150 155 160
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
165 170 175
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
180 185 190
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
195 200 205
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
210 215 220
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
225 230 235 240
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser
245 250 255
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
260 265 270
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
275 280 285
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
290 295 300
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Ala Ser Thr Tyr Arg Val Val
305 310 315 320
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
325 330 335
Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr
340 345 350
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Cys Thr Leu
355 360 365
Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Trp Cys
370 375 380
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
385 390 395 400
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
405 410 415
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser
420 425 430
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
435 440 445
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Gly
450 455 460
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
465 470 475 480
Gly Ser Gln Ala Val Val Thr Gln Glu Ser Ala Leu Thr Thr Ser Pro
485 490 495
Gly Glu Thr Val Thr Leu Thr Cys Arg Ser Ser Thr Gly Ala Val Thr
500 505 510
Thr Ser Asn Tyr Ala Asn Trp Val Gln Glu Lys Pro Asp His Leu Phe
515 520 525
Thr Gly Leu Ile Gly Gly Thr Asn Lys Arg Ala Pro Gly Val Pro Ala
530 535 540
Arg Phe Ser Gly Ser Leu Ile Gly Asp Lys Ala Ala Leu Thr Ile Thr
545 550 555 560
Gly Ala Gln Thr Glu Asp Glu Ala Ile Tyr Phe Cys Ala Leu Trp Tyr
565 570 575
Ser Asn Leu Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu His
580 585 590
His His His His His His His
595
<210> 57
<211> 610
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 57
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys
20 25 30
Pro Gly Ala Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe
35 40 45
Thr Asp Tyr Glu Met His Trp Ile Arg Gln Pro Pro Gly Gln Gly Leu
50 55 60
Glu Trp Ile Gly Ala Ile Asp Pro Lys Thr Gly Asp Thr Ala Tyr Ser
65 70 75 80
Gln Lys Phe Lys Gly Arg Val Thr Leu Thr Ala Asp Lys Ser Thr Ser
85 90 95
Thr Ala Tyr Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Thr Arg Phe Tyr Ser Tyr Thr Tyr Trp Gly Gln Gly Thr
115 120 125
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
130 135 140
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
145 150 155 160
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
165 170 175
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
180 185 190
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
195 200 205
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
210 215 220
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
225 230 235 240
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser
245 250 255
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
260 265 270
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
275 280 285
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
290 295 300
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Ala Ser Thr Tyr Arg Val Val
305 310 315 320
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
325 330 335
Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr
340 345 350
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
355 360 365
Pro Pro Ser Arg Cys Glu Leu Thr Lys Asn Gln Val Ser Leu Ser Cys
370 375 380
Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
385 390 395 400
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
405 410 415
Ser Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val Asp Lys Ser
420 425 430
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
435 440 445
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Gly
450 455 460
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Val Lys
465 470 475 480
Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Lys Gly Ser Leu Lys
485 490 495
Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asn Thr Tyr Ala Met Asn
500 505 510
Trp Val Arg Gln Ala Pro Gly Lys Cys Leu Glu Trp Val Ala Arg Ile
515 520 525
Arg Ser Lys Tyr Asn Asn Tyr Ala Thr Tyr Tyr Ala Asp Ser Val Lys
530 535 540
Asp Arg Phe Thr Ile Ser Arg Asp Asp Ser Gln Ser Ile Leu Tyr Leu
545 550 555 560
Gln Met Asn Asn Leu Lys Thr Glu Asp Thr Ala Met Tyr Tyr Cys Val
565 570 575
Arg His Gly Asn Phe Gly Asn Ser Tyr Val Ser Trp Phe Ala Tyr Trp
580 585 590
Gly Gln Gly Thr Leu Val Thr Val Ser Ala Asp Tyr Lys Asp Asp Asp
595 600 605
Asp Lys
610
<210> 58
<211> 594
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 58
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys
20 25 30
Pro Gly Ala Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe
35 40 45
Thr Asp Tyr Glu Met His Trp Ile Arg Gln Pro Pro Gly Gln Gly Leu
50 55 60
Glu Trp Ile Gly Ala Ile Asp Pro Lys Thr Gly Asp Thr Ala Tyr Ser
65 70 75 80
Gln Lys Phe Lys Gly Arg Val Thr Leu Thr Ala Asp Lys Ser Thr Ser
85 90 95
Thr Ala Tyr Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Thr Arg Phe Tyr Ser Tyr Thr Tyr Trp Gly Gln Gly Thr
115 120 125
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
130 135 140
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
145 150 155 160
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
165 170 175
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
180 185 190
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
195 200 205
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
210 215 220
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
225 230 235 240
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser
245 250 255
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
260 265 270
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
275 280 285
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
290 295 300
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Ala Ser Thr Tyr Arg Val Val
305 310 315 320
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
325 330 335
Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr
340 345 350
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Cys Thr Leu
355 360 365
Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Trp Cys
370 375 380
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
385 390 395 400
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
405 410 415
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser
420 425 430
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
435 440 445
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Gly
450 455 460
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ala Val
465 470 475 480
Val Thr Gln Glu Ser Ala Leu Thr Thr Ser Pro Gly Glu Thr Val Thr
485 490 495
Leu Thr Cys Arg Ser Ser Thr Gly Ala Val Thr Thr Ser Asn Tyr Ala
500 505 510
Asn Trp Val Gln Glu Lys Pro Asp His Leu Phe Thr Gly Leu Ile Gly
515 520 525
Gly Thr Asn Lys Arg Ala Pro Gly Val Pro Ala Arg Phe Ser Gly Ser
530 535 540
Leu Ile Gly Asp Lys Ala Ala Leu Thr Ile Thr Gly Ala Gln Thr Glu
545 550 555 560
Asp Glu Ala Ile Tyr Phe Cys Ala Leu Trp Tyr Ser Asn Leu Trp Val
565 570 575
Phe Gly Cys Gly Thr Lys Leu Thr Val Leu His His His His His His
580 585 590
His His
<210> 59
<211> 704
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 59
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys
20 25 30
Pro Gly Ala Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe
35 40 45
Thr Asp Tyr Glu Met His Trp Ile Arg Gln Pro Pro Gly Gln Gly Leu
50 55 60
Glu Trp Ile Gly Ala Ile Asp Pro Lys Thr Gly Asp Thr Ala Tyr Ser
65 70 75 80
Gln Lys Phe Lys Gly Arg Val Thr Leu Thr Ala Asp Lys Ser Thr Ser
85 90 95
Thr Ala Tyr Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Thr Arg Phe Tyr Ser Tyr Thr Tyr Trp Gly Gln Gly Thr
115 120 125
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
130 135 140
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
145 150 155 160
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
165 170 175
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
180 185 190
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
195 200 205
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
210 215 220
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
225 230 235 240
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser
245 250 255
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
260 265 270
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
275 280 285
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
290 295 300
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Ala Ser Thr Tyr Arg Val Val
305 310 315 320
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
325 330 335
Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr
340 345 350
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
355 360 365
Pro Pro Ser Arg Cys Glu Leu Thr Lys Asn Gln Val Ser Leu Ser Cys
370 375 380
Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
385 390 395 400
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
405 410 415
Ser Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val Asp Lys Ser
420 425 430
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
435 440 445
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Gly
450 455 460
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Val Lys
465 470 475 480
Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Lys Gly Ser Leu Lys
485 490 495
Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asn Thr Tyr Ala Met Asn
500 505 510
Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Arg Ile
515 520 525
Arg Ser Lys Tyr Asn Asn Tyr Ala Thr Tyr Tyr Ala Asp Ser Val Lys
530 535 540
Asp Arg Phe Thr Ile Ser Arg Asp Asp Ser Gln Ser Ile Leu Tyr Leu
545 550 555 560
Gln Met Asn Asn Leu Lys Thr Glu Asp Thr Ala Met Tyr Tyr Cys Val
565 570 575
Arg His Gly Asn Phe Gly Asn Ser Tyr Val Ser Trp Phe Ala Tyr Trp
580 585 590
Gly Gln Gly Thr Leu Val Thr Val Ser Ala Ala Ser Thr Lys Gly Pro
595 600 605
Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr
610 615 620
Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr
625 630 635 640
Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro
645 650 655
Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr
660 665 670
Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn
675 680 685
His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser
690 695 700
<210> 60
<211> 692
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 60
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys
20 25 30
Pro Gly Ala Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe
35 40 45
Thr Asp Tyr Glu Met His Trp Ile Arg Gln Pro Pro Gly Gln Gly Leu
50 55 60
Glu Trp Ile Gly Ala Ile Asp Pro Lys Thr Gly Asp Thr Ala Tyr Ser
65 70 75 80
Gln Lys Phe Lys Gly Arg Val Thr Leu Thr Ala Asp Lys Ser Thr Ser
85 90 95
Thr Ala Tyr Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Thr Arg Phe Tyr Ser Tyr Thr Tyr Trp Gly Gln Gly Thr
115 120 125
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
130 135 140
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
145 150 155 160
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
165 170 175
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
180 185 190
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
195 200 205
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
210 215 220
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
225 230 235 240
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser
245 250 255
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
260 265 270
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
275 280 285
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
290 295 300
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Ala Ser Thr Tyr Arg Val Val
305 310 315 320
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
325 330 335
Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr
340 345 350
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Cys Thr Leu
355 360 365
Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Trp Cys
370 375 380
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
385 390 395 400
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
405 410 415
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser
420 425 430
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
435 440 445
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Gly
450 455 460
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ala Val
465 470 475 480
Val Thr Gln Glu Ser Ala Leu Thr Thr Ser Pro Gly Glu Thr Val Thr
485 490 495
Leu Thr Cys Arg Ser Ser Thr Gly Ala Val Thr Thr Ser Asn Tyr Ala
500 505 510
Asn Trp Val Gln Glu Lys Pro Asp His Leu Phe Thr Gly Leu Ile Gly
515 520 525
Gly Thr Asn Lys Arg Ala Pro Gly Val Pro Ala Arg Phe Ser Gly Ser
530 535 540
Leu Ile Gly Asp Lys Ala Ala Leu Thr Ile Thr Gly Ala Gln Thr Glu
545 550 555 560
Asp Glu Ala Ile Tyr Phe Cys Ala Leu Trp Tyr Ser Asn Leu Trp Val
565 570 575
Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Arg Thr Val Ala Ala Pro
580 585 590
Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr
595 600 605
Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys
610 615 620
Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu
625 630 635 640
Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser
645 650 655
Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala
660 665 670
Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe
675 680 685
Asn Arg Gly Glu
690
<210> 61
<211> 115
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 61
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Glu Met His Trp Ile Arg Gln Pro Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Ala Ile Asp Pro Lys Thr Gly Asp Thr Ala Tyr Ser Gln Lys Phe
50 55 60
Lys Gly Arg Val Thr Leu Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Arg Phe Tyr Ser Tyr Thr Tyr Trp Gly Gln Gly Thr Leu Val Thr
100 105 110
Val Ser Ser
115
<210> 62
<211> 219
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 62
Asp Val Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Val His Ser
20 25 30
Asn Arg Asn Thr Tyr Leu His Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Ser Gln Asn
85 90 95
Thr His Val Pro Pro Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105 110
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
115 120 125
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
130 135 140
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
145 150 155 160
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
165 170 175
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
180 185 190
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
195 200 205
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 63
<211> 328
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 63
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys
1 5 10 15
Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr
65 70 75 80
Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys
100 105 110
Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro
115 120 125
Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys
130 135 140
Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp
145 150 155 160
Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu
165 170 175
Glu Gln Tyr Ala Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu
180 185 190
His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn
195 200 205
Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly
210 215 220
Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu
225 230 235 240
Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr
245 250 255
Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn
260 265 270
Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe
275 280 285
Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn
290 295 300
Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr
305 310 315 320
Gln Lys Ser Leu Ser Leu Ser Pro
325
<210> 64
<211> 587
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 64
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys
1 5 10 15
Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr
65 70 75 80
Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys
100 105 110
Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro
115 120 125
Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys
130 135 140
Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp
145 150 155 160
Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu
165 170 175
Glu Gln Tyr Ala Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu
180 185 190
His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn
195 200 205
Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly
210 215 220
Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu
225 230 235 240
Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr
245 250 255
Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn
260 265 270
Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe
275 280 285
Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn
290 295 300
Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr
305 310 315 320
Gln Lys Ser Leu Ser Leu Ser Pro Gly Gly Gly Gly Ser Gly Gly Gly
325 330 335
Gly Ser Gln Ala Val Val Thr Gln Glu Ser Ala Leu Thr Thr Ser Pro
340 345 350
Gly Glu Thr Val Thr Leu Thr Cys Arg Ser Ser Thr Gly Ala Val Thr
355 360 365
Thr Ser Asn Tyr Ala Asn Trp Val Gln Glu Lys Pro Asp His Leu Phe
370 375 380
Thr Gly Leu Ile Gly Gly Thr Asn Lys Arg Ala Pro Gly Val Pro Ala
385 390 395 400
Arg Phe Ser Gly Ser Leu Ile Gly Asp Lys Ala Ala Leu Thr Ile Thr
405 410 415
Gly Ala Gln Thr Glu Asp Glu Ala Ile Tyr Phe Cys Ala Leu Trp Tyr
420 425 430
Ser Asn Leu Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly
435 440 445
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Val
450 455 460
Lys Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Lys Gly Ser Leu
465 470 475 480
Lys Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asn Thr Tyr Ala Met
485 490 495
Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Arg
500 505 510
Ile Arg Ser Lys Tyr Asn Asn Tyr Ala Thr Tyr Tyr Ala Asp Ser Val
515 520 525
Lys Asp Arg Phe Thr Ile Ser Arg Asp Asp Ser Gln Ser Ile Leu Tyr
530 535 540
Leu Gln Met Asn Asn Leu Lys Thr Glu Asp Thr Ala Met Tyr Tyr Cys
545 550 555 560
Val Arg His Gly Asn Phe Gly Asn Ser Tyr Val Ser Trp Phe Ala Tyr
565 570 575
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ala
580 585
<210> 65
<211> 328
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 65
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys
1 5 10 15
Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr
65 70 75 80
Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys
100 105 110
Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro
115 120 125
Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys
130 135 140
Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp
145 150 155 160
Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu
165 170 175
Glu Gln Tyr Ala Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu
180 185 190
His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn
195 200 205
Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly
210 215 220
Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu
225 230 235 240
Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr
245 250 255
Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn
260 265 270
Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe
275 280 285
Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn
290 295 300
Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn Arg Tyr Thr
305 310 315 320
Gln Glu Ser Leu Ser Leu Ser Pro
325
<210> 66
<211> 587
<212> PRT
<213> Искусственная
<220>
<223> Искусственно синтезированная последовательность
<400> 66
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys
1 5 10 15
Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr
65 70 75 80
Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys
100 105 110
Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro
115 120 125
Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys
130 135 140
Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp
145 150 155 160
Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu
165 170 175
Glu Gln Tyr Ala Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu
180 185 190
His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn
195 200 205
Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly
210 215 220
Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Lys Glu
225 230 235 240
Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr
245 250 255
Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn
260 265 270
Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe
275 280 285
Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn
290 295 300
Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr
305 310 315 320
Gln Lys Ser Leu Ser Leu Ser Pro Gly Gly Gly Gly Ser Gly Gly Gly
325 330 335
Gly Ser Gln Ala Val Val Thr Gln Glu Ser Ala Leu Thr Thr Ser Pro
340 345 350
Gly Glu Thr Val Thr Leu Thr Cys Arg Ser Ser Thr Gly Ala Val Thr
355 360 365
Thr Ser Asn Tyr Ala Asn Trp Val Gln Glu Lys Pro Asp His Leu Phe
370 375 380
Thr Gly Leu Ile Gly Gly Thr Asn Lys Arg Ala Pro Gly Val Pro Ala
385 390 395 400
Arg Phe Ser Gly Ser Leu Ile Gly Asp Lys Ala Ala Leu Thr Ile Thr
405 410 415
Gly Ala Gln Thr Glu Asp Glu Ala Ile Tyr Phe Cys Ala Leu Trp Tyr
420 425 430
Ser Asn Leu Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly
435 440 445
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Val
450 455 460
Lys Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Lys Gly Ser Leu
465 470 475 480
Lys Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asn Thr Tyr Ala Met
485 490 495
Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Arg
500 505 510
Ile Arg Ser Lys Tyr Asn Asn Tyr Ala Thr Tyr Tyr Ala Asp Ser Val
515 520 525
Lys Asp Arg Phe Thr Ile Ser Arg Asp Asp Ser Gln Ser Ile Leu Tyr
530 535 540
Leu Gln Met Asn Asn Leu Lys Thr Glu Asp Thr Ala Met Tyr Tyr Cys
545 550 555 560
Val Arg His Gly Asn Phe Gly Asn Ser Tyr Val Ser Trp Phe Ala Tyr
565 570 575
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ala
580 585
<210> 67
<211> 141
<212> PRT
<213> Homo sapiens
<400> 67
Asp Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys
1 5 10 15
Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr
20 25 30
Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr
35 40 45
Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala
50 55 60
Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser
65 70 75 80
Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp
85 90 95
Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe
100 105 110
Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala
115 120 125
Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
130 135 140
<210> 68
<211> 179
<212> PRT
<213> Homo sapiens
<400> 68
Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu Pro
1 5 10 15
Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys Leu
20 25 30
Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val Asn
35 40 45
Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro Leu Lys
50 55 60
Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg Leu
65 70 75 80
Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg Cys
85 90 95
Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln Asp
100 105 110
Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly Arg
115 120 125
Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu Ser
130 135 140
Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr Ala
145 150 155 160
Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys Asp
165 170 175
Ser Arg Gly
<210> 69
<211> 173
<212> PRT
<213> Homo sapiens
<400> 69
Asp Lys Gln Leu Asp Ala Asp Val Ser Pro Lys Pro Thr Ile Phe Leu
1 5 10 15
Pro Ser Ile Ala Glu Thr Lys Leu Gln Lys Ala Gly Thr Tyr Leu Cys
20 25 30
Leu Leu Glu Lys Phe Phe Pro Asp Val Ile Lys Ile His Trp Gln Glu
35 40 45
Lys Lys Ser Asn Thr Ile Leu Gly Ser Gln Glu Gly Asn Thr Met Lys
50 55 60
Thr Asn Asp Thr Tyr Met Lys Phe Ser Trp Leu Thr Val Pro Glu Lys
65 70 75 80
Ser Leu Asp Lys Glu His Arg Cys Ile Val Arg His Glu Asn Asn Lys
85 90 95
Asn Gly Val Asp Gln Glu Ile Ile Phe Pro Pro Ile Lys Thr Asp Val
100 105 110
Ile Thr Met Asp Pro Lys Asp Asn Cys Ser Lys Asp Ala Asn Asp Thr
115 120 125
Leu Leu Leu Gln Leu Thr Asn Thr Ser Ala Tyr Tyr Met Tyr Leu Leu
130 135 140
Leu Leu Leu Lys Ser Val Val Tyr Phe Ala Ile Ile Thr Cys Cys Leu
145 150 155 160
Leu Arg Arg Thr Ala Phe Cys Cys Asn Gly Glu Lys Ser
165 170
<210> 70
<211> 204
<212> PRT
<213> Homo sapiens
<400> 70
Lys Gln Leu Asp Ala Asp Val Ser Pro Lys Pro Thr Ile Phe Leu Pro
1 5 10 15
Ser Ile Ala Glu Thr Lys Leu Gln Lys Ala Gly Thr Tyr Leu Cys Leu
20 25 30
Leu Glu Lys Phe Phe Pro Asp Ile Ile Lys Ile His Trp Gln Glu Lys
35 40 45
Lys Ser Asn Thr Ile Leu Gly Ser Gln Glu Gly Asn Thr Met Lys Thr
50 55 60
Asn Asp Thr Tyr Met Lys Phe Ser Trp Leu Thr Val Pro Glu Glu Ser
65 70 75 80
Leu Asp Lys Glu His Arg Cys Ile Val Arg His Glu Asn Asn Lys Asn
85 90 95
Gly Ile Asp Gln Glu Ile Ile Phe Pro Pro Ile Lys Thr Asp Val Thr
100 105 110
Thr Val Asp Pro Lys Asp Ser Tyr Ser Lys Asp Ala Asn Asp Val Thr
115 120 125
Thr Val Asp Pro Lys Tyr Asn Tyr Ser Lys Asp Ala Asn Asp Val Ile
130 135 140
Thr Met Asp Pro Lys Asp Asn Trp Ser Lys Asp Ala Asn Asp Thr Leu
145 150 155 160
Leu Leu Gln Leu Thr Asn Thr Ser Ala Tyr Tyr Met Tyr Leu Leu Leu
165 170 175
Leu Leu Lys Ser Val Val Tyr Phe Ala Ile Ile Thr Cys Cys Leu Leu
180 185 190
Gly Arg Thr Ala Phe Cys Cys Asn Gly Glu Lys Ser
195 200
<210> 71
<211> 177
<212> PRT
<213> Homo sapiens
<400> 71
Pro Ser Tyr Thr Gly Gly Tyr Ala Asp Lys Leu Ile Phe Gly Lys Gly
1 5 10 15
Thr Arg Val Thr Val Glu Pro Arg Ser Gln Pro His Thr Lys Pro Ser
20 25 30
Val Phe Val Met Lys Asn Gly Thr Asn Val Ala Cys Leu Val Lys Glu
35 40 45
Phe Tyr Pro Lys Asp Ile Arg Ile Asn Leu Val Ser Ser Lys Lys Ile
50 55 60
Thr Glu Phe Asp Pro Ala Ile Val Ile Ser Pro Ser Gly Lys Tyr Asn
65 70 75 80
Ala Val Lys Leu Gly Lys Tyr Glu Asp Ser Asn Ser Val Thr Cys Ser
85 90 95
Val Gln His Asp Asn Lys Thr Val His Ser Thr Asp Phe Glu Val Lys
100 105 110
Thr Asp Ser Thr Asp His Val Lys Pro Lys Glu Thr Glu Asn Thr Lys
115 120 125
Gln Pro Ser Lys Ser Cys His Lys Pro Lys Ala Ile Val His Thr Glu
130 135 140
Lys Val Asn Met Met Ser Leu Thr Val Leu Gly Leu Arg Met Leu Phe
145 150 155 160
Ala Lys Thr Val Ala Val Asn Phe Leu Leu Thr Ala Lys Leu Phe Phe
165 170 175
Leu
<210> 72
<211> 19
<212> PRT
<213> Искусственная
<220>
<223> сигнальная последовательность
<400> 72
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser
<210> 73
<211> 466
<212> PRT
<213> Искусственная последовательность
<220>
<223> Искусственно синтезированная последовательность
<400> 73
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Ala Val Val Thr Gln Glu Ser Ala Leu Thr Thr Ser
20 25 30
Pro Gly Glu Thr Val Thr Leu Thr Cys Arg Ser Ser Thr Gly Ala Val
35 40 45
Thr Thr Ser Asn Tyr Ala Asn Trp Val Gln Glu Lys Pro Asp His Leu
50 55 60
Phe Thr Gly Leu Ile Gly Gly Thr Asn Lys Arg Ala Pro Gly Val Pro
65 70 75 80
Ala Arg Phe Ser Gly Ser Leu Ile Gly Asp Lys Ala Ala Leu Thr Ile
85 90 95
Thr Gly Ala Gln Thr Glu Asp Glu Ala Ile Tyr Phe Cys Ala Leu Trp
100 105 110
Tyr Ser Asn Leu Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
115 120 125
Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser
130 135 140
Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys
145 150 155 160
Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu
165 170 175
Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu
180 185 190
Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr
195 200 205
Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val
210 215 220
Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro
225 230 235 240
Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe
245 250 255
Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val
260 265 270
Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe
275 280 285
Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro
290 295 300
Arg Glu Glu Gln Tyr Ala Ser Thr Tyr Arg Val Val Ser Val Leu Thr
305 310 315 320
Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val
325 330 335
Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala
340 345 350
Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg
355 360 365
Cys Glu Leu Thr Lys Asn Gln Val Ser Leu Ser Cys Ala Val Lys Gly
370 375 380
Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro
385 390 395 400
Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser
405 410 415
Phe Phe Leu Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln
420 425 430
Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His
435 440 445
Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Asp Tyr Lys Asp Asp Asp
450 455 460
Asp Lys
465
<210> 74
<211> 251
<212> PRT
<213> Искусственная последовательность
<220>
<223> Искусственно синтезированная последовательность
<400> 74
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Glu Val Lys Leu Leu Glu Ser Gly Gly Gly Leu Val Gln
20 25 30
Pro Lys Gly Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
35 40 45
Asn Thr Tyr Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
Glu Trp Val Ala Arg Ile Arg Ser Lys Tyr Asn Asn Tyr Ala Thr Tyr
65 70 75 80
Tyr Ala Asp Ser Val Lys Asp Arg Phe Thr Ile Ser Arg Asp Asp Ser
85 90 95
Gln Ser Ile Leu Tyr Leu Gln Met Asn Asn Leu Lys Thr Glu Asp Thr
100 105 110
Ala Met Tyr Tyr Cys Val Arg His Gly Asn Phe Gly Asn Ser Tyr Val
115 120 125
Ser Trp Phe Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ala
130 135 140
Ala Ser Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
145 150 155 160
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
165 170 175
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
180 185 190
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
195 200 205
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
210 215 220
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
225 230 235 240
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
245 250
<210> 75
<211> 484
<212> PRT
<213> Искусственная последовательность
<220>
<223> Искусственно синтезированная последовательность
<400> 75
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Glu Val Lys Leu Leu Glu Ser Gly Gly Gly Leu Val Gln
20 25 30
Pro Lys Gly Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
35 40 45
Asn Thr Tyr Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
Glu Trp Val Ala Arg Ile Arg Ser Lys Tyr Asn Asn Tyr Ala Thr Tyr
65 70 75 80
Tyr Ala Asp Ser Val Lys Asp Arg Phe Thr Ile Ser Arg Asp Asp Ser
85 90 95
Gln Ser Ile Leu Tyr Leu Gln Met Asn Asn Leu Lys Thr Glu Asp Thr
100 105 110
Ala Met Tyr Tyr Cys Val Arg His Gly Asn Phe Gly Asn Ser Tyr Val
115 120 125
Ser Trp Phe Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ala
130 135 140
Ala Ser Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
145 150 155 160
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
165 170 175
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
180 185 190
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
195 200 205
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
210 215 220
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
225 230 235 240
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys Asp Lys Thr His Thr
245 250 255
Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe
260 265 270
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
275 280 285
Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val
290 295 300
Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
305 310 315 320
Lys Pro Arg Glu Glu Gln Tyr Ala Ser Thr Tyr Arg Val Val Ser Val
325 330 335
Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
340 345 350
Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser
355 360 365
Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro
370 375 380
Ser Arg Cys Glu Leu Thr Lys Asn Gln Val Ser Leu Ser Cys Ala Val
385 390 395 400
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
405 410 415
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp
420 425 430
Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
435 440 445
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
450 455 460
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Asp Tyr Lys Asp
465 470 475 480
Asp Asp Asp Lys
<210> 76
<211> 233
<212> PRT
<213> Искусственная последовательность
<220>
<223> Искусственно синтезированная последовательность
<400> 76
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Ala Val Val Thr Gln Glu Ser Ala Leu Thr Thr Ser
20 25 30
Pro Gly Glu Thr Val Thr Leu Thr Cys Arg Ser Ser Thr Gly Ala Val
35 40 45
Thr Thr Ser Asn Tyr Ala Asn Trp Val Gln Glu Lys Pro Asp His Leu
50 55 60
Phe Thr Gly Leu Ile Gly Gly Thr Asn Lys Arg Ala Pro Gly Val Pro
65 70 75 80
Ala Arg Phe Ser Gly Ser Leu Ile Gly Asp Lys Ala Ala Leu Thr Ile
85 90 95
Thr Gly Ala Gln Thr Glu Asp Glu Ala Ile Tyr Phe Cys Ala Leu Trp
100 105 110
Tyr Ser Asn Leu Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
115 120 125
Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser
130 135 140
Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys
145 150 155 160
Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu
165 170 175
Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu
180 185 190
Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr
195 200 205
Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val
210 215 220
Asp Lys Lys Val Glu Pro Lys Ser Cys
225 230
<210> 77
<211> 466
<212> PRT
<213> Искусственная последовательность
<220>
<223> Искусственно синтезированная последовательность
<400> 77
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Thr Val Val Thr Gln Glu Pro Ser Leu Thr Val Ser
20 25 30
Pro Gly Gly Thr Val Thr Leu Thr Cys Gly Ser Ser Thr Gly Ala Val
35 40 45
Thr Ser Gly Asn Tyr Pro Asn Trp Val Gln Gln Lys Pro Gly Gln Ala
50 55 60
Pro Arg Gly Leu Ile Gly Gly Thr Lys Phe Leu Ala Pro Gly Thr Pro
65 70 75 80
Ala Arg Phe Ser Gly Ser Leu Leu Gly Gly Lys Ala Ala Leu Thr Leu
85 90 95
Ser Gly Val Gln Pro Glu Asp Glu Ala Glu Tyr Tyr Cys Val Leu Trp
100 105 110
Tyr Ser Asn Arg Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
115 120 125
Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser
130 135 140
Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys
145 150 155 160
Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu
165 170 175
Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu
180 185 190
Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr
195 200 205
Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val
210 215 220
Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro
225 230 235 240
Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe
245 250 255
Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val
260 265 270
Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe
275 280 285
Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro
290 295 300
Arg Glu Glu Gln Tyr Ala Ser Thr Tyr Arg Val Val Ser Val Leu Thr
305 310 315 320
Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val
325 330 335
Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala
340 345 350
Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg
355 360 365
Cys Glu Leu Thr Lys Asn Gln Val Ser Leu Ser Cys Ala Val Lys Gly
370 375 380
Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro
385 390 395 400
Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser
405 410 415
Phe Phe Leu Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln
420 425 430
Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His
435 440 445
Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Asp Tyr Lys Asp Asp Asp
450 455 460
Asp Lys
465
<210> 78
<211> 251
<212> PRT
<213> Искусственная последовательность
<220>
<223> Искусственно синтезированная последовательность
<400> 78
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln
20 25 30
Pro Gly Gly Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
35 40 45
Asn Lys Tyr Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
Glu Trp Val Ala Arg Ile Arg Ser Lys Tyr Asn Asn Tyr Ala Thr Tyr
65 70 75 80
Tyr Ala Asp Ser Val Lys Asp Arg Phe Thr Ile Ser Arg Asp Asp Ser
85 90 95
Lys Asn Thr Ala Tyr Leu Gln Met Asn Asn Leu Lys Thr Glu Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Val Arg His Gly Asn Phe Gly Asn Ser Tyr Ile
115 120 125
Ser Tyr Trp Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
130 135 140
Ala Ser Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
145 150 155 160
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
165 170 175
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
180 185 190
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
195 200 205
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
210 215 220
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
225 230 235 240
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
245 250
<210> 79
<211> 475
<212> PRT
<213> Искусственная последовательность
<220>
<223> Искусственно синтезированная последовательность
<400> 79
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Glu Val Gln Leu Leu Glu Gln Ser Gly Ala Glu Leu Val
20 25 30
Arg Pro Gly Thr Ser Val Lys Ile Ser Cys Lys Ala Ser Gly Tyr Ala
35 40 45
Phe Thr Asn Tyr Trp Leu Gly Trp Val Lys Gln Arg Pro Gly His Gly
50 55 60
Leu Glu Trp Ile Gly Asp Ile Phe Pro Gly Ser Gly Asn Ile His Tyr
65 70 75 80
Asn Glu Lys Phe Lys Gly Lys Ala Thr Leu Thr Ala Asp Lys Ser Ser
85 90 95
Ser Thr Ala Tyr Met Gln Leu Ser Ser Leu Thr Phe Glu Asp Ser Ala
100 105 110
Val Tyr Phe Cys Ala Arg Leu Arg Asn Trp Asp Glu Pro Met Asp Tyr
115 120 125
Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly
130 135 140
Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly
145 150 155 160
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
165 170 175
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
180 185 190
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
195 200 205
Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val
210 215 220
Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys
225 230 235 240
Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala
245 250 255
Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
260 265 270
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
275 280 285
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
290 295 300
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Ala Ser
305 310 315 320
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
325 330 335
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala
340 345 350
Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
355 360 365
Gln Val Cys Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln
370 375 380
Val Ser Leu Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
385 390 395 400
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
405 410 415
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
420 425 430
Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser
435 440 445
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
450 455 460
Leu Ser Pro His His His His His His His His
465 470 475
<210> 80
<211> 239
<212> PRT
<213> Искусственная последовательность
<220>
<223> Искусственно синтезированная последовательность
<400> 80
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Glu Leu Val Met Thr Gln Ser Pro Ser Ser Leu Thr Val
20 25 30
Thr Ala Gly Glu Lys Val Thr Met Ser Cys Lys Ser Ser Gln Ser Leu
35 40 45
Leu Asn Ser Gly Asn Gln Lys Asn Tyr Leu Thr Trp Tyr Gln Gln Lys
50 55 60
Pro Gly Gln Pro Pro Lys Leu Leu Ile Tyr Trp Ala Ser Thr Arg Glu
65 70 75 80
Ser Gly Val Pro Asp Arg Phe Thr Gly Ser Gly Ser Gly Thr Asp Phe
85 90 95
Thr Leu Thr Ile Ser Ser Val Gln Ala Glu Asp Leu Ala Val Tyr Tyr
100 105 110
Cys Gln Asn Asp Tyr Ser Tyr Pro Leu Thr Phe Gly Ala Gly Thr Lys
115 120 125
Leu Glu Ile Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro
130 135 140
Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu
145 150 155 160
Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp
165 170 175
Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp
180 185 190
Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys
195 200 205
Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln
210 215 220
Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230 235
<210> 81
<211> 453
<212> PRT
<213> Искусственная последовательность
<220>
<223> Искусственно синтезированная последовательность
<400> 81
Glu Val Lys Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Lys Gly
1 5 10 15
Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asn Thr Tyr
20 25 30
Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Arg Ile Arg Ser Lys Tyr Asn Asn Tyr Ala Thr Tyr Tyr Ala Asp
50 55 60
Ser Val Lys Asp Arg Phe Thr Ile Ser Arg Asp Asp Ser Gln Ser Ile
65 70 75 80
Leu Tyr Leu Gln Met Asn Asn Leu Lys Thr Glu Asp Thr Ala Met Tyr
85 90 95
Tyr Cys Val Arg His Gly Asn Phe Gly Asn Ser Tyr Val Ser Trp Phe
100 105 110
Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ala Ala Ser Thr
115 120 125
Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser
130 135 140
Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu
145 150 155 160
Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His
165 170 175
Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser
180 185 190
Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys
195 200 205
Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu
210 215 220
Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro
225 230 235 240
Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
245 250 255
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
260 265 270
Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp
275 280 285
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr
290 295 300
Ala Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
305 310 315 320
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu
325 330 335
Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
340 345 350
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys
355 360 365
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
370 375 380
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
385 390 395 400
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
405 410 415
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser
420 425 430
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
435 440 445
Leu Ser Leu Ser Pro
450
<210> 82
<211> 216
<212> PRT
<213> Искусственная последовательность
<220>
<223> Искусственно синтезированная последовательность
<400> 82
Gln Ala Val Val Thr Gln Glu Ser Ala Leu Thr Thr Ser Pro Gly Glu
1 5 10 15
Thr Val Thr Leu Thr Cys Arg Ser Ser Thr Gly Ala Val Thr Thr Ser
20 25 30
Asn Tyr Ala Asn Trp Val Gln Glu Lys Pro Asp His Leu Phe Thr Gly
35 40 45
Leu Ile Gly Gly Thr Asn Lys Arg Ala Pro Gly Val Pro Ala Arg Phe
50 55 60
Ser Gly Ser Leu Ile Gly Asp Lys Ala Ala Leu Thr Ile Thr Gly Ala
65 70 75 80
Gln Thr Glu Asp Glu Ala Ile Tyr Phe Cys Ala Leu Trp Tyr Ser Asn
85 90 95
Leu Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Arg Thr Val
100 105 110
Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys
115 120 125
Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg
130 135 140
Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn
145 150 155 160
Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser
165 170 175
Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys
180 185 190
Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr
195 200 205
Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 83
<211> 443
<212> PRT
<213> Искусственная последовательность
<220>
<223> Искусственно синтезированная последовательность
<400> 83
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Glu Met His Trp Ile Arg Gln Pro Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Ala Ile Asp Pro Lys Thr Gly Asp Thr Ala Tyr Ser Gln Lys Phe
50 55 60
Lys Gly Arg Val Thr Leu Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Arg Phe Tyr Ser Tyr Thr Tyr Trp Gly Gln Gly Thr Leu Val Thr
100 105 110
Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro
115 120 125
Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val
130 135 140
Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala
145 150 155 160
Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly
165 170 175
Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly
180 185 190
Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys
195 200 205
Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys
210 215 220
Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu
225 230 235 240
Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu
245 250 255
Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys
260 265 270
Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys
275 280 285
Pro Arg Glu Glu Gln Tyr Ala Ser Thr Tyr Arg Val Val Ser Val Leu
290 295 300
Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys
305 310 315 320
Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys
325 330 335
Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser
340 345 350
Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys
355 360 365
Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln
370 375 380
Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly
385 390 395 400
Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln
405 410 415
Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn
420 425 430
His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440
<210> 84
<211> 219
<212> PRT
<213> Искусственная последовательность
<220>
<223> Искусственно синтезированная последовательность
<400> 84
Asp Ile Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Val His Ser
20 25 30
Asn Arg Asn Thr Tyr Leu His Trp Tyr Gln Gln Lys Pro Gly Gln Ala
35 40 45
Pro Arg Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Ser Gln Asn
85 90 95
Thr His Val Pro Pro Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105 110
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
115 120 125
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
130 135 140
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
145 150 155 160
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
165 170 175
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
180 185 190
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
195 200 205
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 85
<211> 453
<212> PRT
<213> Искусственная последовательность
<220>
<223> Искусственно синтезированная последовательность
<400> 85
Glu Val Lys Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Lys Gly
1 5 10 15
Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asn Thr Tyr
20 25 30
Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Arg Ile Arg Ser Lys Tyr Asn Asn Tyr Ala Thr Tyr Tyr Ala Asp
50 55 60
Ser Val Lys Asp Arg Phe Thr Ile Ser Arg Asp Asp Ser Gln Ser Ile
65 70 75 80
Leu Tyr Leu Gln Met Asn Asn Leu Lys Thr Glu Asp Thr Ala Met Tyr
85 90 95
Tyr Cys Val Arg His Gly Asn Phe Gly Asn Ser Tyr Val Ser Trp Phe
100 105 110
Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ala Ala Ser Thr
115 120 125
Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser
130 135 140
Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu
145 150 155 160
Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His
165 170 175
Thr Phe Pro Ala Val Leu Lys Ser Ser Gly Leu Tyr Ser Leu Ser Ser
180 185 190
Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys
195 200 205
Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu
210 215 220
Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro
225 230 235 240
Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
245 250 255
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
260 265 270
Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp
275 280 285
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr
290 295 300
Ala Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
305 310 315 320
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu
325 330 335
Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
340 345 350
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Cys Glu Leu Thr Lys
355 360 365
Asn Gln Val Ser Leu Ser Cys Ala Val Lys Gly Phe Tyr Pro Ser Asp
370 375 380
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
385 390 395 400
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Val Ser
405 410 415
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser
420 425 430
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
435 440 445
Leu Ser Leu Ser Pro
450
<210> 86
<211> 216
<212> PRT
<213> Искусственная последовательность
<220>
<223> Искусственно синтезированная последовательность
<400> 86
Gln Ala Val Val Thr Gln Glu Ser Ala Leu Thr Thr Ser Pro Gly Glu
1 5 10 15
Thr Val Thr Leu Thr Cys Arg Ser Ser Thr Gly Ala Val Thr Thr Ser
20 25 30
Asn Tyr Ala Asn Trp Val Gln Glu Lys Pro Asp His Leu Phe Thr Gly
35 40 45
Leu Ile Gly Gly Thr Asn Lys Arg Ala Pro Gly Val Pro Ala Arg Phe
50 55 60
Ser Gly Ser Leu Ile Gly Asp Lys Ala Ala Leu Thr Ile Thr Gly Ala
65 70 75 80
Gln Thr Glu Asp Glu Ala Ile Tyr Phe Cys Ala Leu Trp Tyr Ser Asn
85 90 95
Leu Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Arg Thr Val
100 105 110
Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys
115 120 125
Ser Gly Thr Ala Glu Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg
130 135 140
Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn
145 150 155 160
Ser Glu Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser
165 170 175
Leu Ser Ser Thr Leu Glu Leu Ser Lys Ala Asp Tyr Glu Lys His Lys
180 185 190
Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr
195 200 205
Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 87
<211> 443
<212> PRT
<213> Искусственная последовательность
<220>
<223> Искусственно синтезированная последовательность
<400> 87
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Glu Met His Trp Ile Arg Gln Pro Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Ala Ile Asp Pro Lys Thr Gly Asp Thr Ala Tyr Ser Gln Lys Phe
50 55 60
Lys Gly Arg Val Thr Leu Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Arg Phe Tyr Ser Tyr Thr Tyr Trp Gly Gln Gly Thr Leu Val Thr
100 105 110
Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro
115 120 125
Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val
130 135 140
Glu Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala
145 150 155 160
Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Glu Ser Ser Gly
165 170 175
Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly
180 185 190
Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys
195 200 205
Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys
210 215 220
Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu
225 230 235 240
Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu
245 250 255
Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys
260 265 270
Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys
275 280 285
Pro Arg Glu Glu Gln Tyr Ala Ser Thr Tyr Arg Val Val Ser Val Leu
290 295 300
Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys
305 310 315 320
Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys
325 330 335
Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Cys Thr Leu Pro Pro Ser
340 345 350
Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Trp Cys Leu Val Lys
355 360 365
Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln
370 375 380
Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly
385 390 395 400
Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln
405 410 415
Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn
420 425 430
His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440
<210> 88
<211> 443
<212> PRT
<213> Искусственная последовательность
<220>
<223> Искусственно синтезированная последовательность
<400> 88
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Glu Met His Trp Ile Arg Gln Pro Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Ala Ile Asp Pro Lys Thr Gly Asp Thr Ala Tyr Ser Gln Lys Phe
50 55 60
Lys Gly Arg Val Thr Leu Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Arg Phe Tyr Ser Tyr Thr Tyr Trp Gly Gln Gly Thr Leu Val Thr
100 105 110
Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro
115 120 125
Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val
130 135 140
Glu Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala
145 150 155 160
Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Glu Ser Ser Gly
165 170 175
Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly
180 185 190
Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys
195 200 205
Val Asp Glu Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys
210 215 220
Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu
225 230 235 240
Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu
245 250 255
Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys
260 265 270
Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys
275 280 285
Pro Arg Glu Glu Gln Tyr Ala Ser Thr Tyr Arg Val Val Ser Val Leu
290 295 300
Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys
305 310 315 320
Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys
325 330 335
Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Cys Thr Leu Pro Pro Ser
340 345 350
Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Trp Cys Leu Val Lys
355 360 365
Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln
370 375 380
Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly
385 390 395 400
Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln
405 410 415
Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn
420 425 430
His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440
<210> 89
<211> 219
<212> PRT
<213> Искусственная последовательность
<220>
<223> Искусственно синтезированная последовательность
<400> 89
Asp Ile Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Val His Ser
20 25 30
Asn Arg Asn Thr Tyr Leu His Trp Tyr Gln Gln Lys Pro Gly Gln Ala
35 40 45
Pro Arg Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Ser Gln Asn
85 90 95
Thr His Val Pro Pro Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105 110
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
115 120 125
Gln Leu Lys Ser Gly Thr Ala Lys Val Val Cys Leu Leu Asn Asn Phe
130 135 140
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
145 150 155 160
Ser Gly Asn Ser Lys Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
165 170 175
Thr Tyr Ser Leu Ser Ser Thr Leu Lys Leu Ser Lys Ala Asp Tyr Glu
180 185 190
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
195 200 205
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 90
<211> 219
<212> PRT
<213> Искусственная последовательность
<220>
<223> Искусственно синтезированная последовательность
<400> 90
Asp Ile Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Val His Ser
20 25 30
Asn Arg Asn Thr Tyr Leu His Trp Tyr Gln Gln Lys Pro Gly Gln Ala
35 40 45
Pro Arg Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Ser Gln Asn
85 90 95
Thr His Val Pro Pro Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105 110
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Lys
115 120 125
Gln Leu Lys Ser Gly Thr Ala Lys Val Val Cys Leu Leu Asn Asn Phe
130 135 140
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
145 150 155 160
Ser Gly Asn Ser Lys Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
165 170 175
Thr Tyr Ser Leu Ser Ser Thr Leu Lys Leu Ser Lys Ala Asp Tyr Glu
180 185 190
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
195 200 205
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 91
<211> 474
<212> PRT
<213> Искусственная последовательность
<220>
<223> Искусственно синтезированная последовательность
<400> 91
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Lys Gln Ser Gly Pro Gly Leu Val Gln
20 25 30
Pro Ser Gln Ser Leu Ser Ile Thr Cys Thr Val Ser Gly Phe Ser Leu
35 40 45
Thr Asn Tyr Gly Val His Trp Val Arg Gln Ser Pro Gly Lys Gly Leu
50 55 60
Glu Trp Leu Gly Val Ile Trp Ser Gly Gly Asn Thr Asp Tyr Asn Thr
65 70 75 80
Pro Phe Thr Ser Arg Leu Ser Ile Asn Lys Asp Asn Ser Lys Ser Gln
85 90 95
Val Phe Phe Lys Met Asn Ser Leu Gln Ser Asn Asp Thr Ala Ile Tyr
100 105 110
Tyr Cys Ala Arg Ala Leu Thr Tyr Tyr Asp Tyr Glu Phe Ala Tyr Trp
115 120 125
Gly Gln Gly Thr Leu Val Thr Val Ser Ala Ala Ser Thr Lys Gly Pro
130 135 140
Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr
145 150 155 160
Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr
165 170 175
Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro
180 185 190
Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr
195 200 205
Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn
210 215 220
His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser
225 230 235 240
Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala
245 250 255
Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu
260 265 270
Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser
275 280 285
His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu
290 295 300
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Ala Ser Thr
305 310 315 320
Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn
325 330 335
Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro
340 345 350
Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln
355 360 365
Val Cys Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val
370 375 380
Ser Leu Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val
385 390 395 400
Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro
405 410 415
Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr
420 425 430
Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val
435 440 445
Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu
450 455 460
Ser Pro His His His His His His His His
465 470
<210> 92
<211> 233
<212> PRT
<213> Искусственная последовательность
<220>
<223> Искусственно синтезированная последовательность
<400> 92
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Asp Ile Leu Leu Thr Gln Ser Pro Val Ile Leu Ser Val
20 25 30
Ser Pro Gly Glu Arg Val Ser Phe Ser Cys Arg Ala Ser Gln Ser Ile
35 40 45
Gly Thr Asn Ile His Trp Tyr Gln Gln Arg Thr Asn Gly Ser Pro Arg
50 55 60
Leu Leu Ile Lys Tyr Ala Ser Glu Ser Ile Ser Gly Ile Pro Ser Arg
65 70 75 80
Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Ser Ile Asn Ser
85 90 95
Val Glu Ser Glu Asp Ile Ala Asp Tyr Tyr Cys Gln Gln Asn Asn Asn
100 105 110
Trp Pro Thr Thr Phe Gly Ala Gly Thr Lys Leu Glu Leu Lys Arg Thr
115 120 125
Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu
130 135 140
Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro
145 150 155 160
Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly
165 170 175
Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr
180 185 190
Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His
195 200 205
Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val
210 215 220
Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| НОВЫЕ СЛИТЫЕ БЕЛКИ, СПЕЦИФИЧЕСКИЕ В ОТНОШЕНИИ CD137 И GPC3 | 2020 |
|
RU2814653C2 |
| БЕЛОК, СВЯЗЫВАЮЩИЙСЯ С NKG2D, CD16 И С ОПУХОЛЕСПЕЦИФИЧЕСКИМ АНТИГЕНОМ | 2018 |
|
RU2788531C2 |
| ИММУНОАКТИВИРУЮЩАЯ АНТИГЕНСВЯЗЫВАЮЩАЯ МОЛЕКУЛА | 2015 |
|
RU2722788C2 |
| СЛИТЫЙ ПОЛИПЕПТИД С ПРОТИВОРАКОВОЙ АКТИВНОСТЬЮ | 2016 |
|
RU2754466C2 |
| ИНДУЦИРУЮЩИЙ ЦИТОТОКСИЧНОСТЬ ТЕРАПЕВТИЧЕСКИЙ АГЕНТ | 2015 |
|
RU2743464C2 |
| МУТЕИНЫ ЛИПОКАЛИНА 2 ЧЕЛОВЕКА С АФФИННОСТЬЮ В ОТНОШЕНИИ ГЛИПИКАНА-3 (GPC3) | 2016 |
|
RU2756318C2 |
| БИСПЕЦИФИЧЕСКИЕ АНТИТЕЛА, СВЯЗЫВАЮЩИЕСЯ С TFR | 2019 |
|
RU2810756C2 |
| ТРИСПЕЦИФИЧЕСКИЕ СВЯЗЫВАЮЩИЕ БЕЛКИ, ОТНОСЯЩИЕСЯ К НИМ СПОСОБЫ И ВАРИАНТЫ ИХ ПРИМЕНЕНИЯ | 2020 |
|
RU2822200C2 |
| ТРИСПЕЦИФИЧЕСКИЕ И/ИЛИ ТРИВАЛЕНТНЫЕ СВЯЗЫВАЮЩИЕ БЕЛКИ С ПРИМЕНЕНИЕМ КРОССОВЕРНОГО ФОРМАТА С ДВОЙНЫМ ВАРИАБЕЛЬНЫМ ДОМЕНОМ (CODV) ДЛЯ ЛЕЧЕНИЯ ИНФЕКЦИИ, ВЫЗВАННОЙ HIV | 2020 |
|
RU2820164C2 |
| СЛИТЫЕ СЕРПИНОВЫЕ ПОЛИПЕПТИДЫ И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2019 |
|
RU2728861C1 |

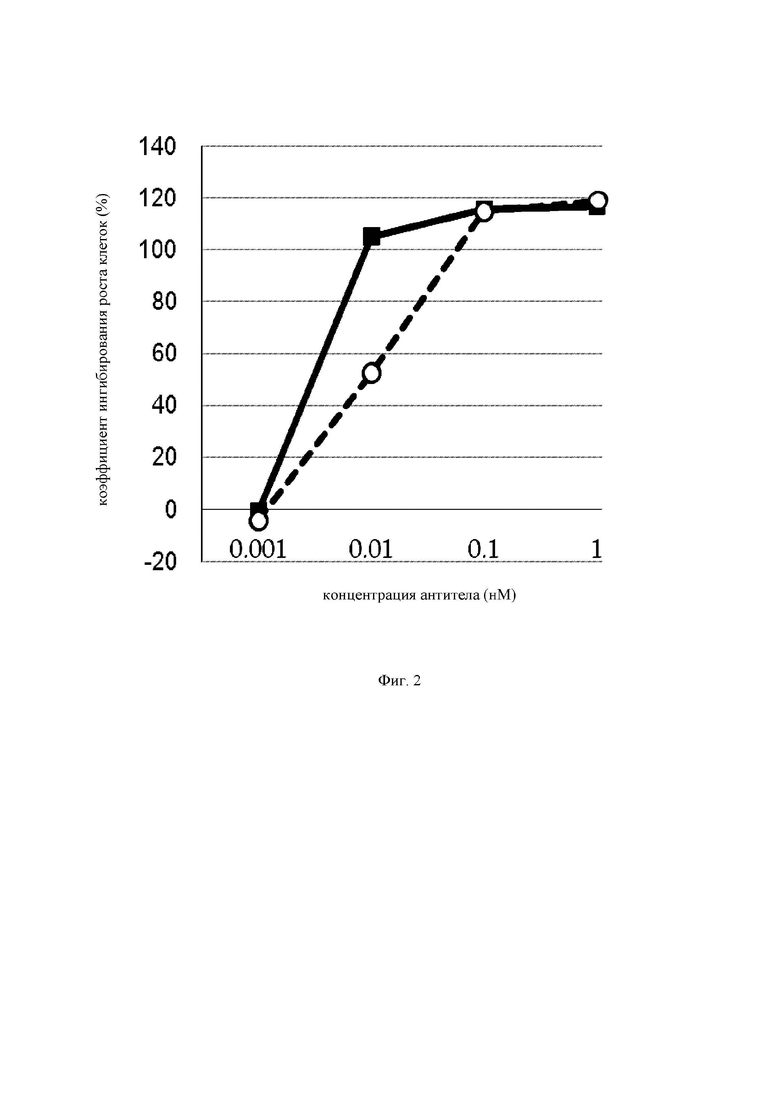
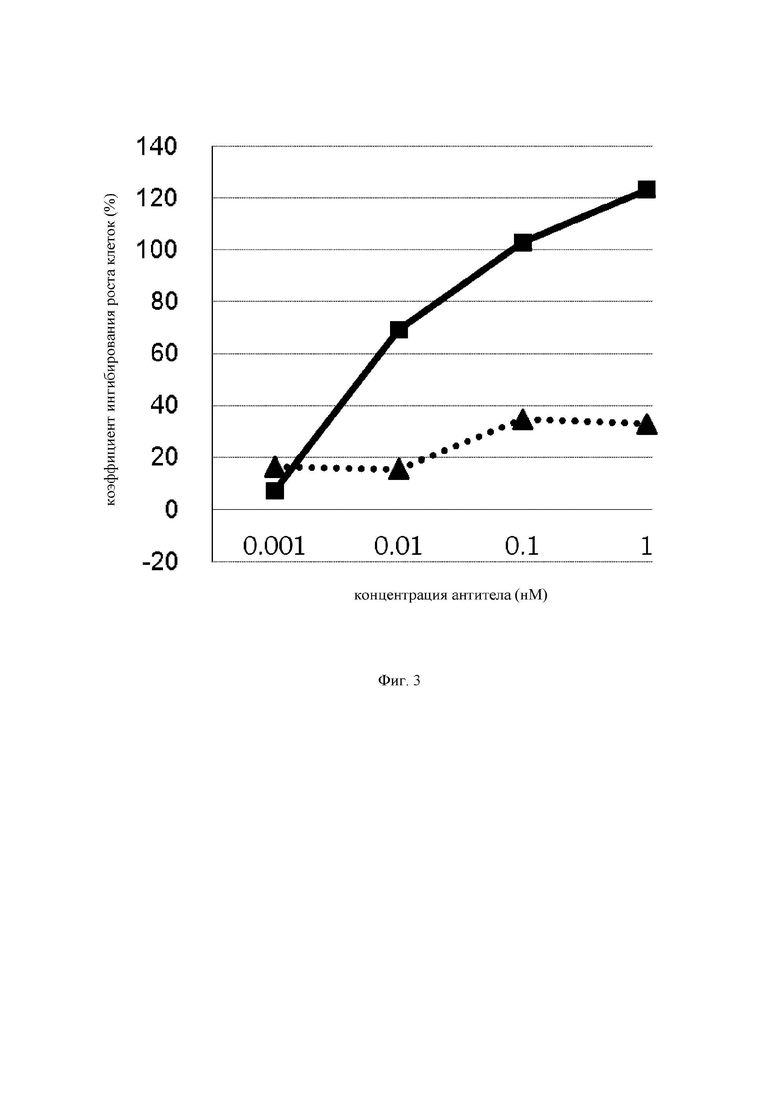
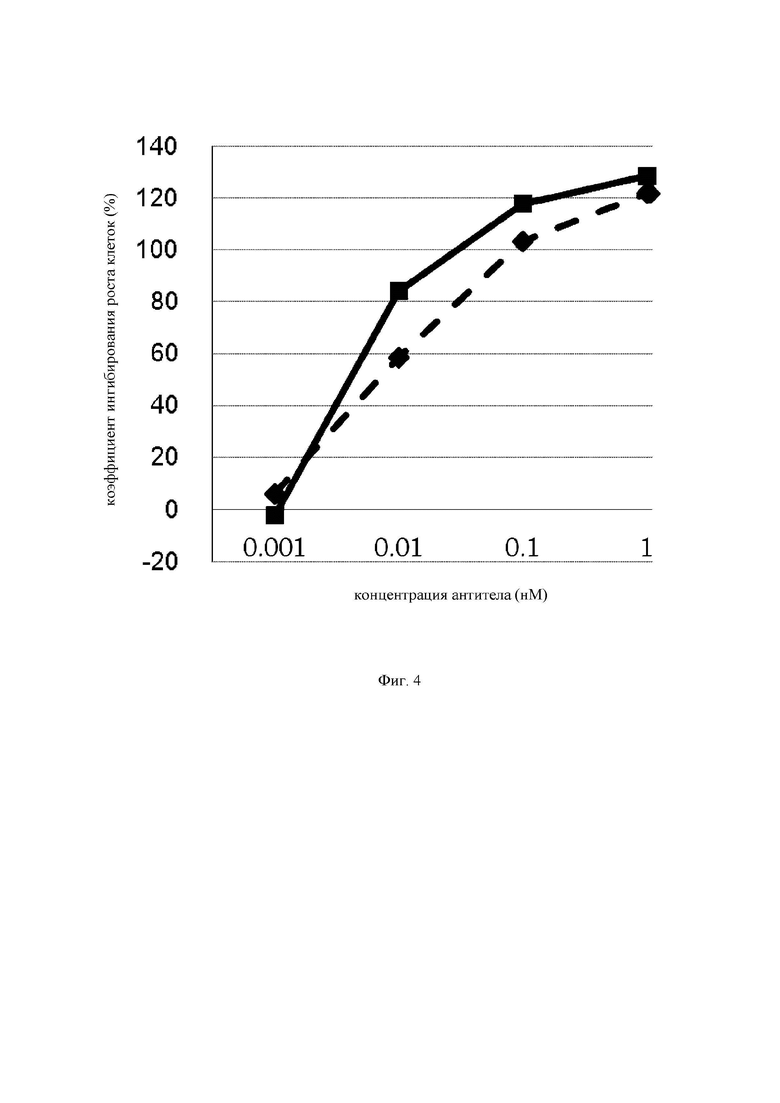
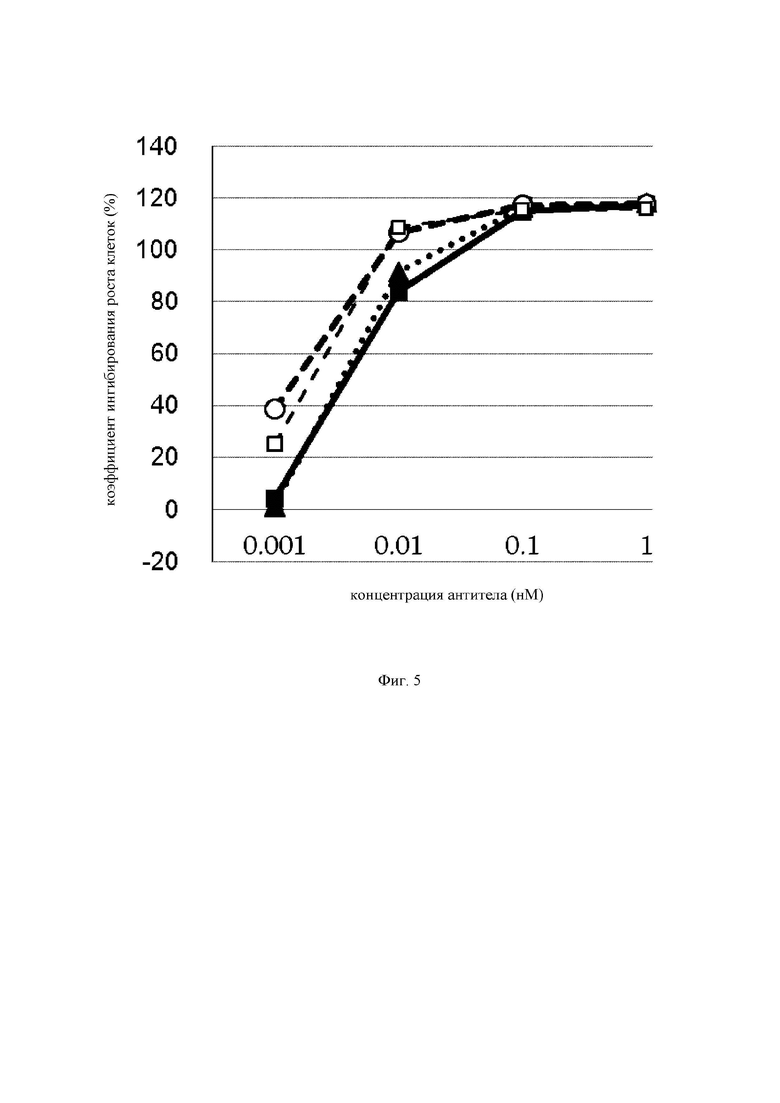
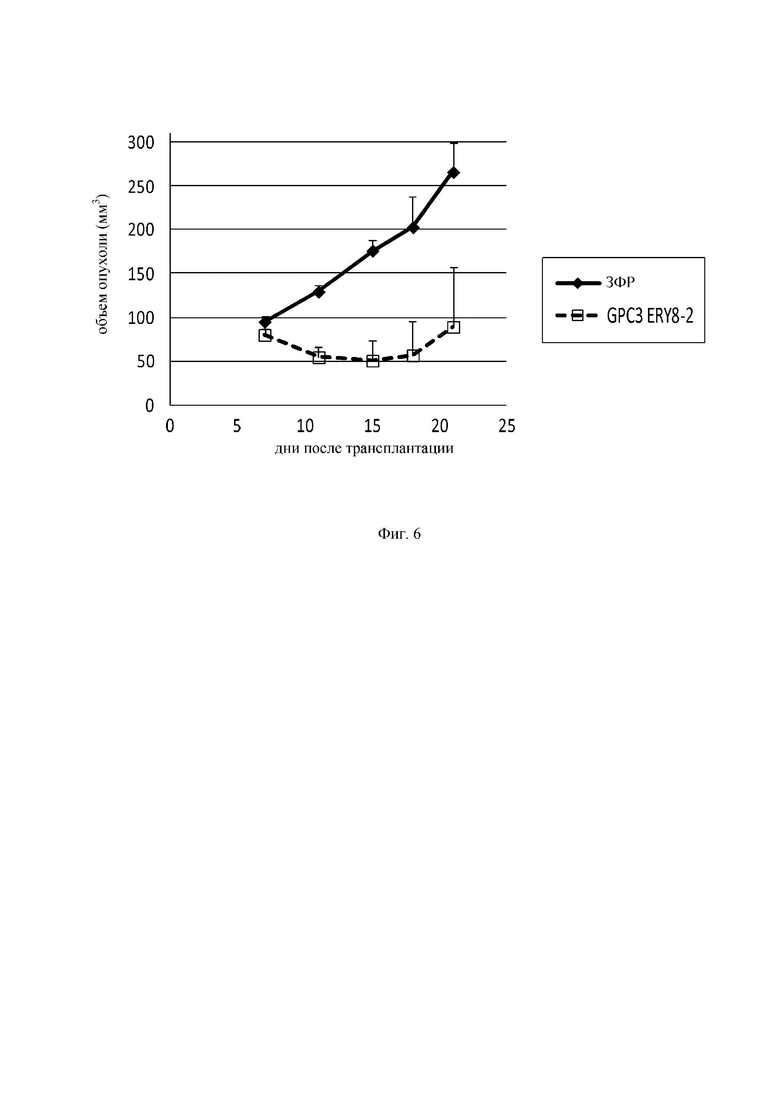
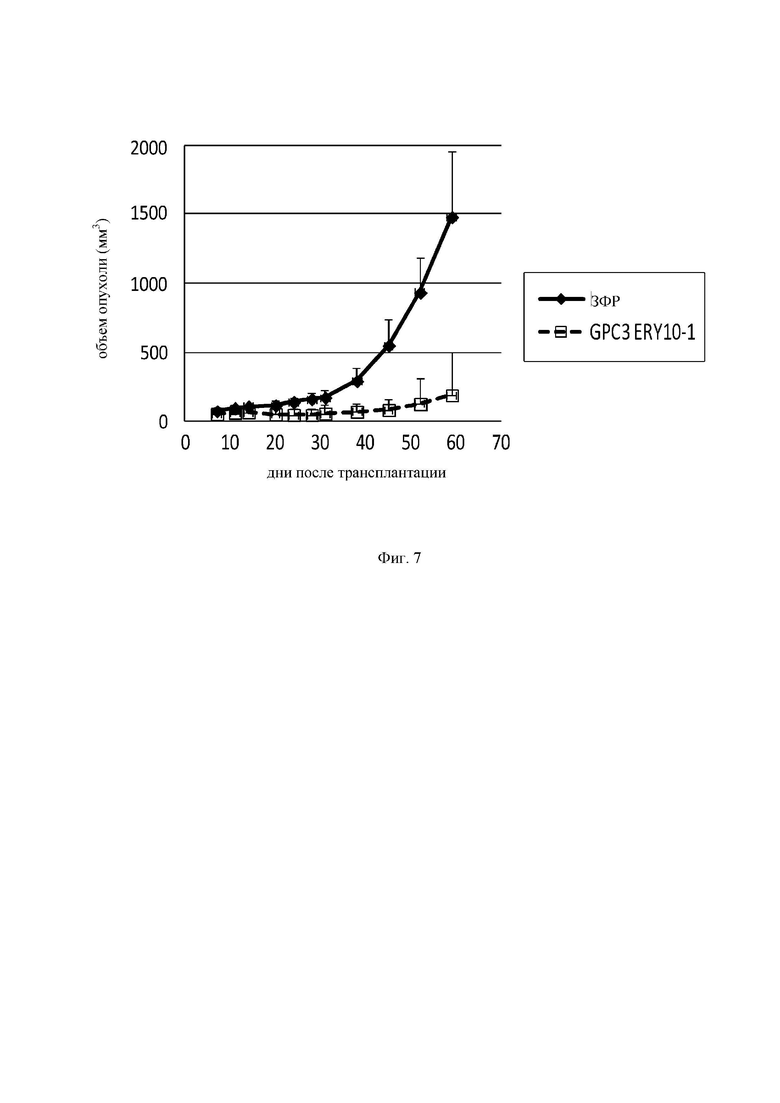
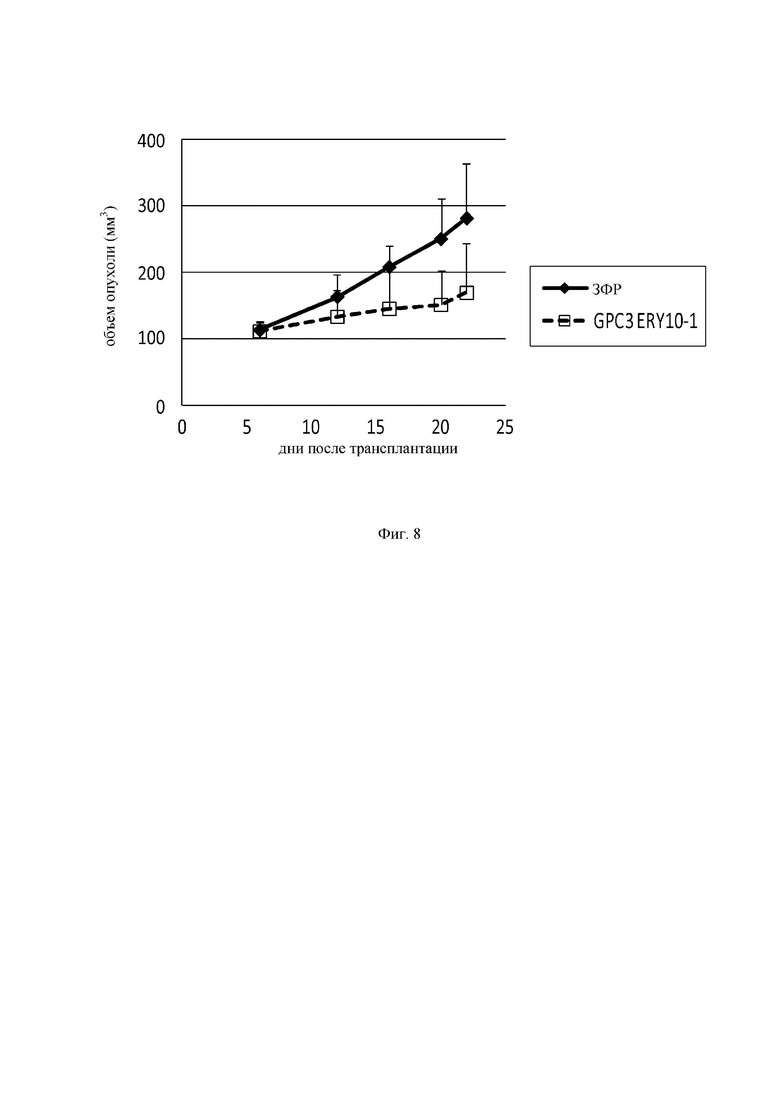
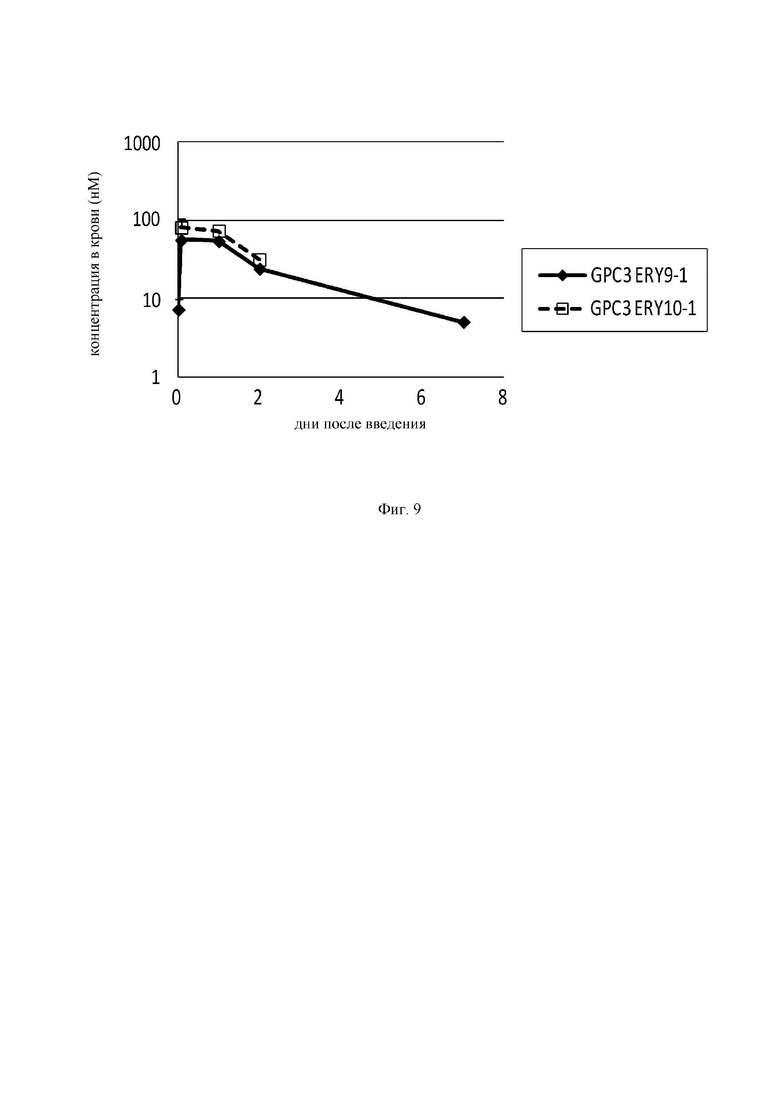
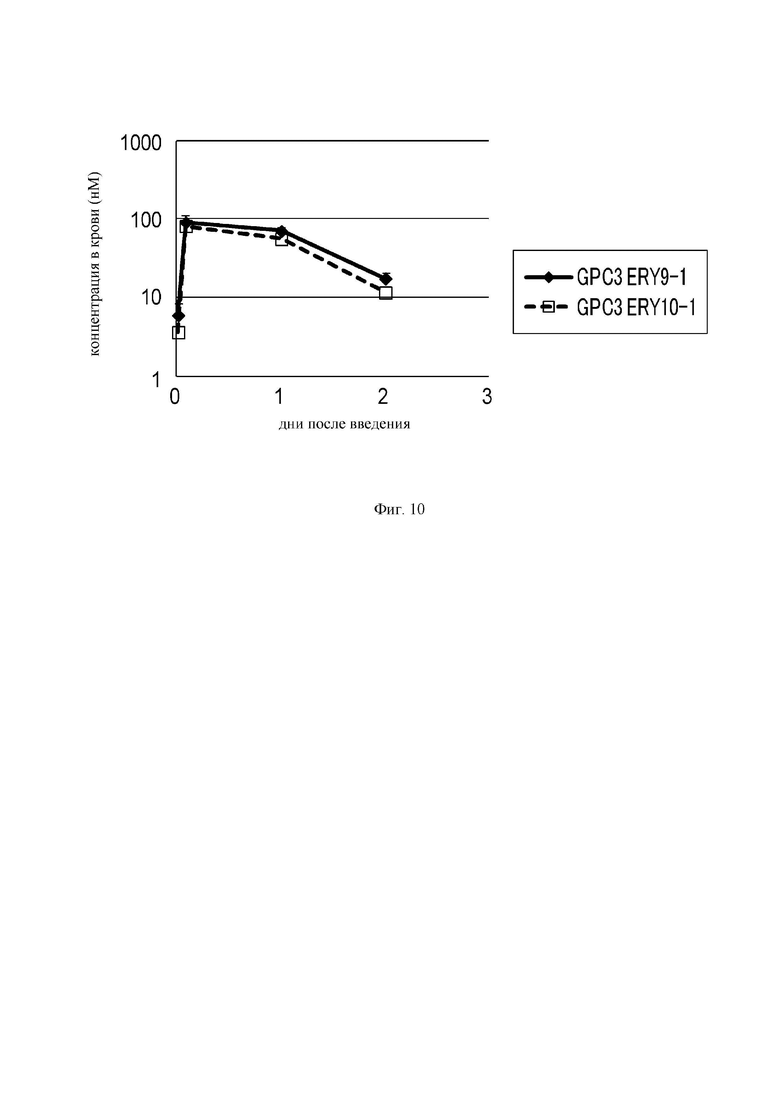
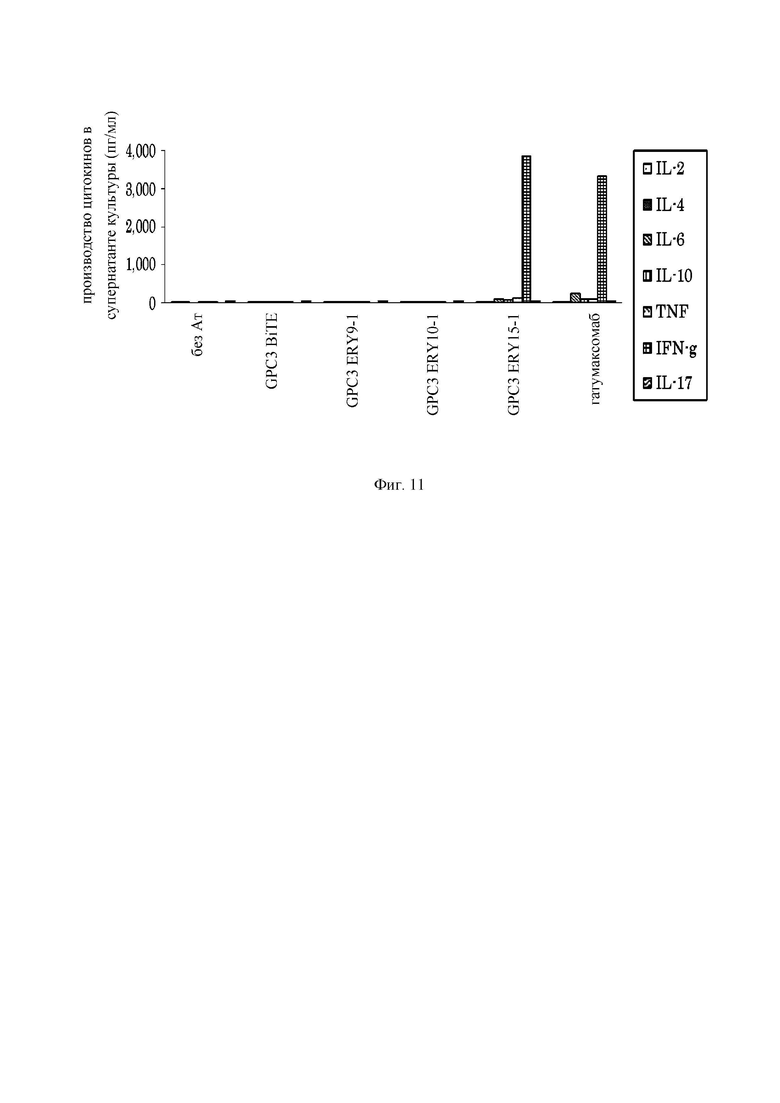
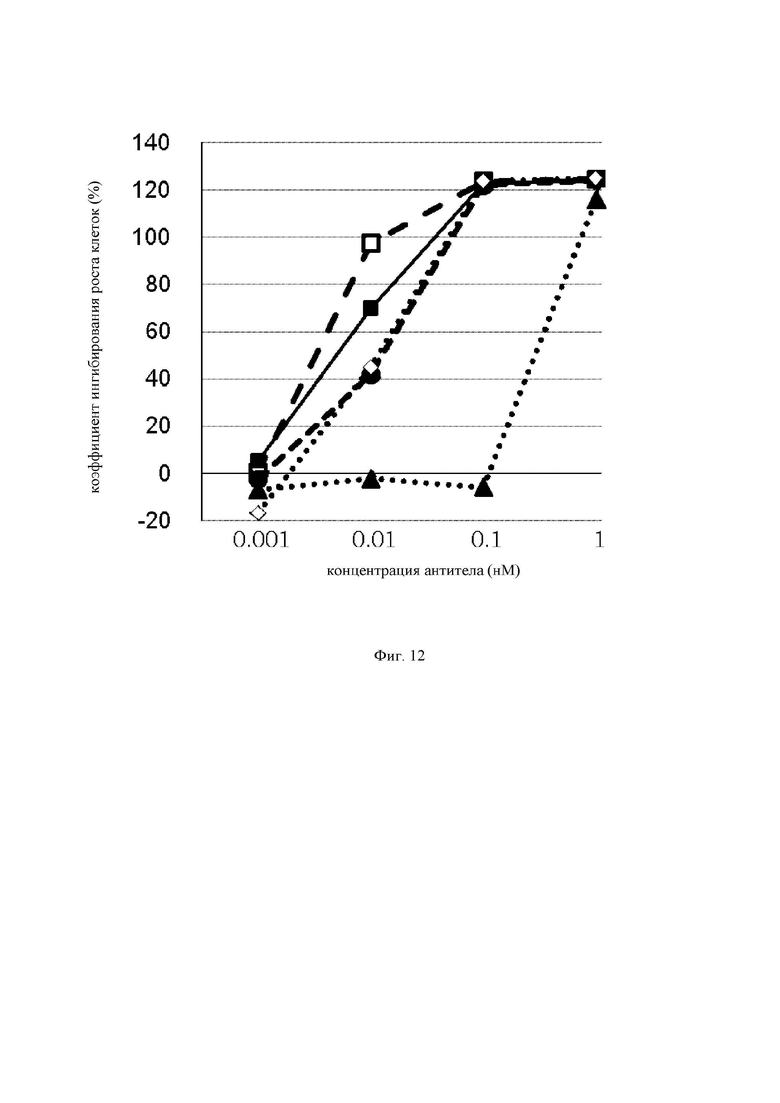
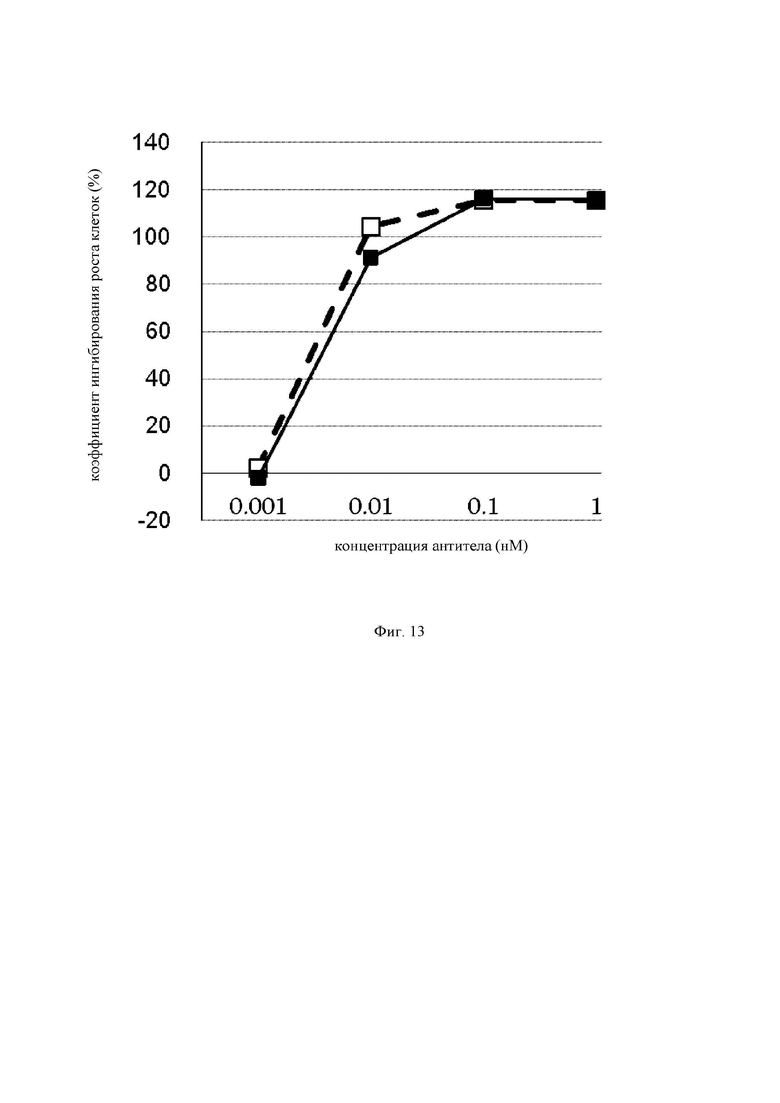
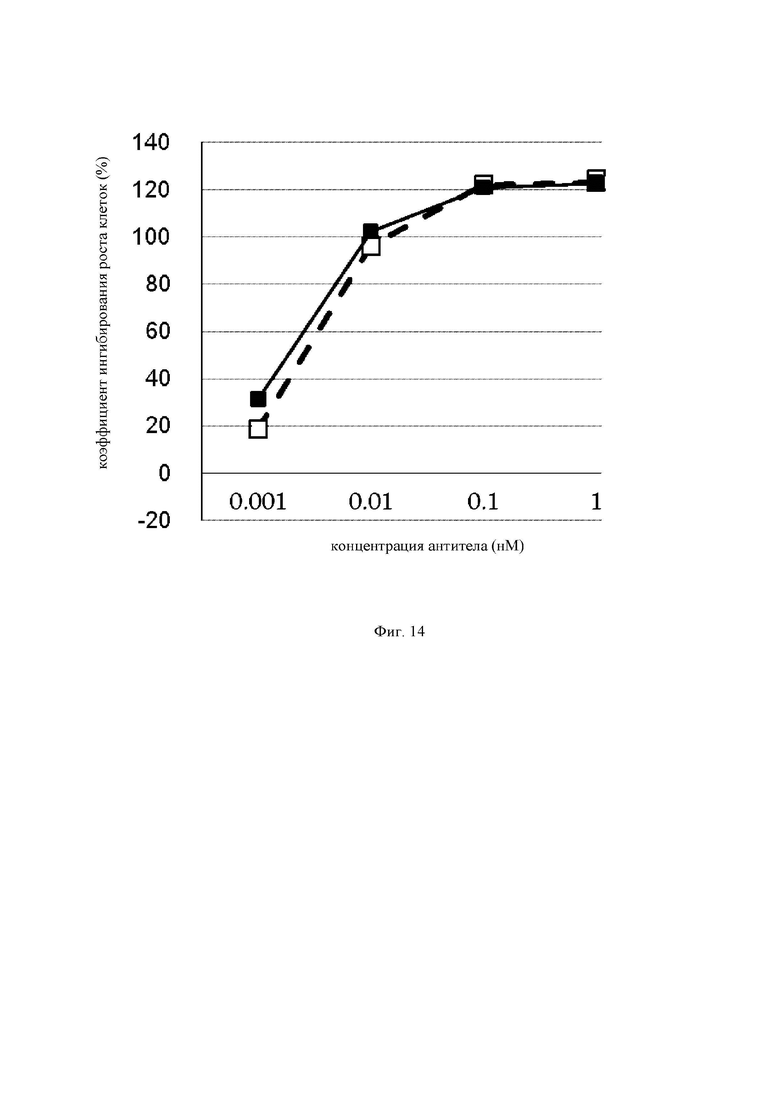
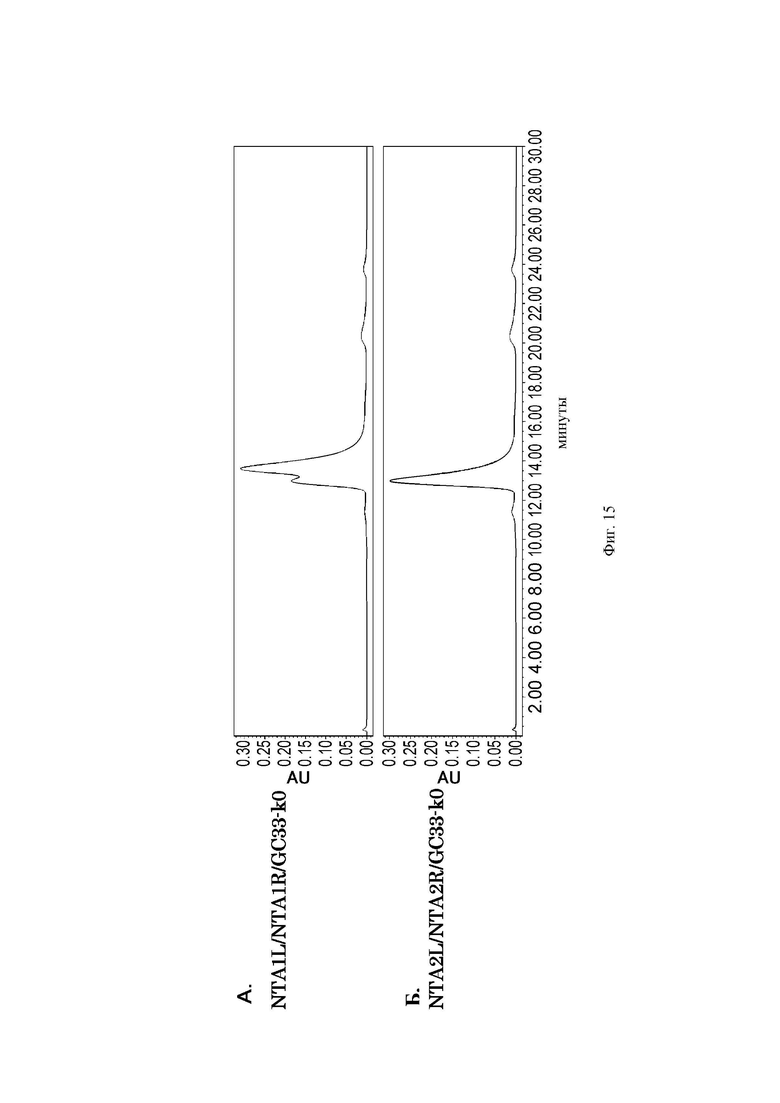

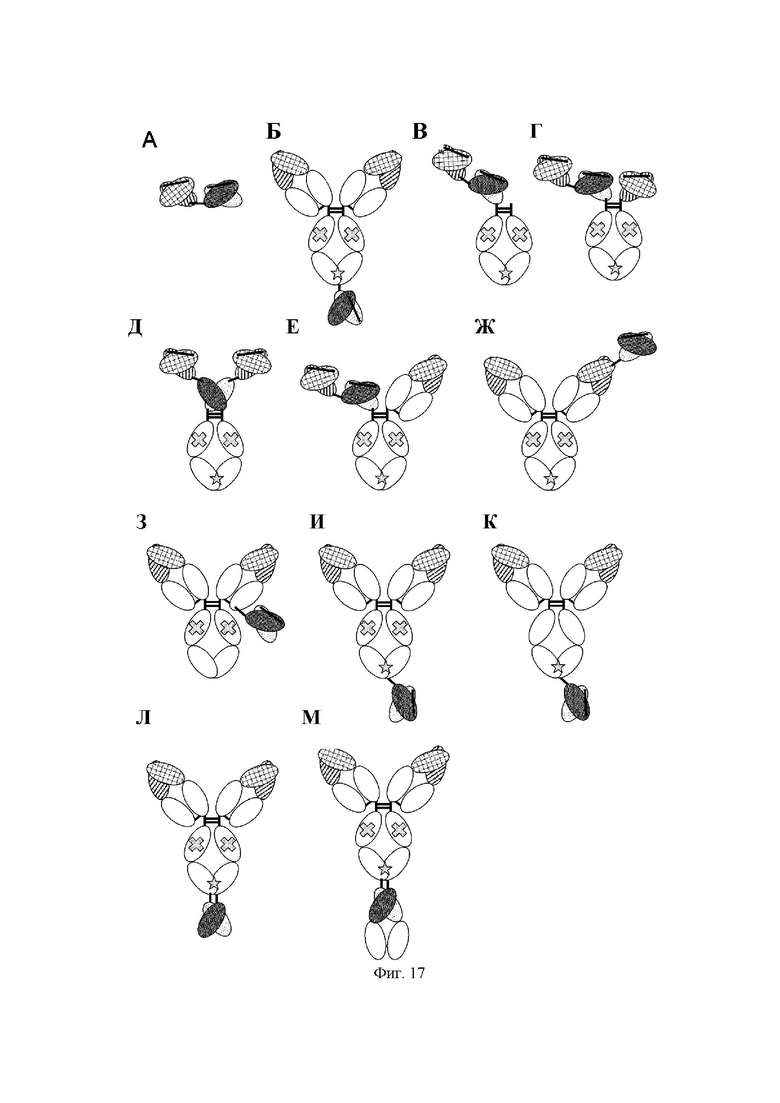
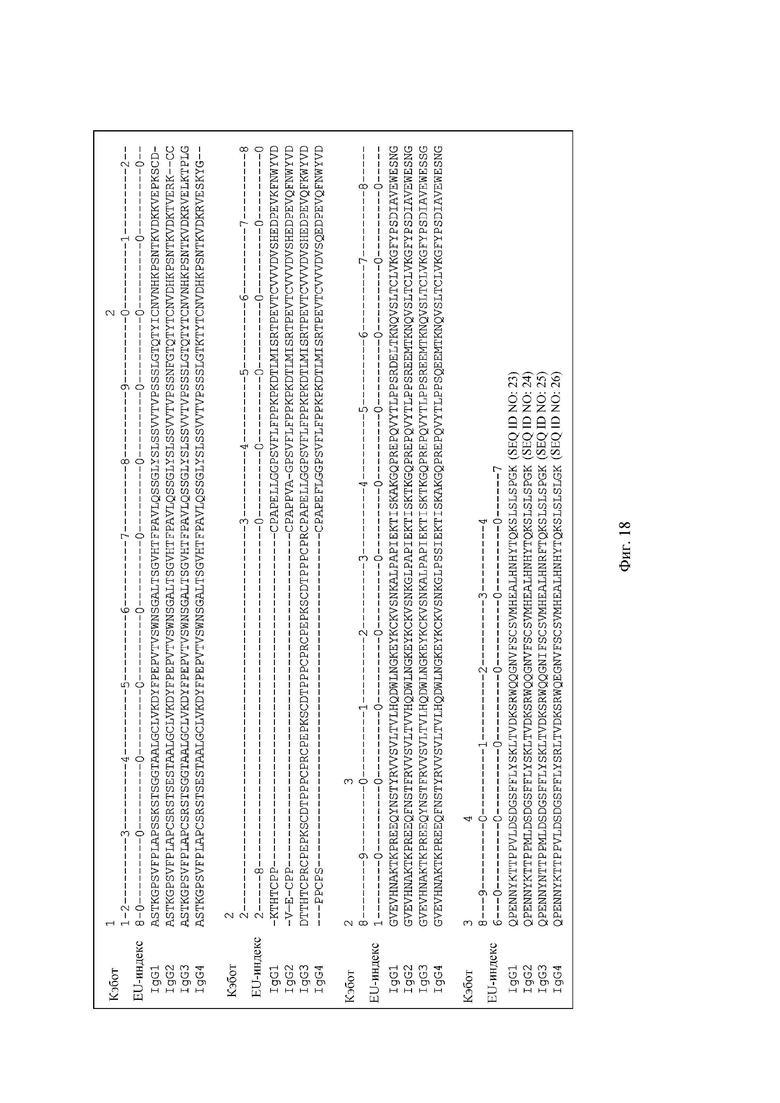
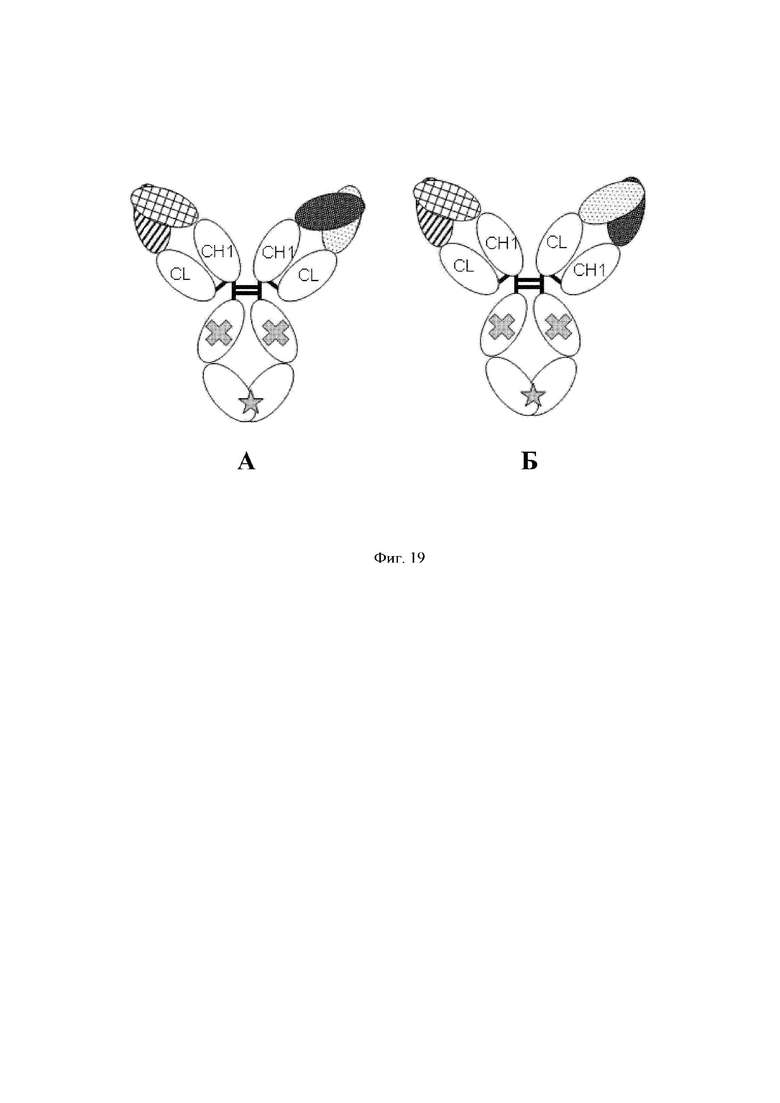
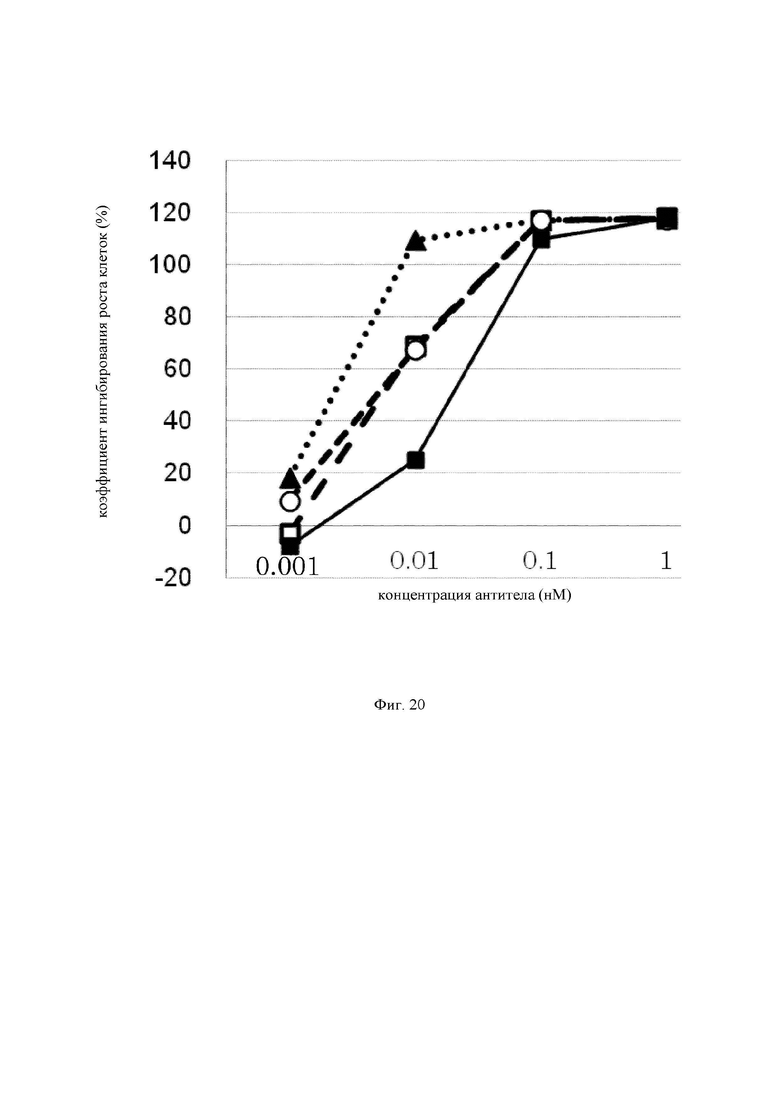
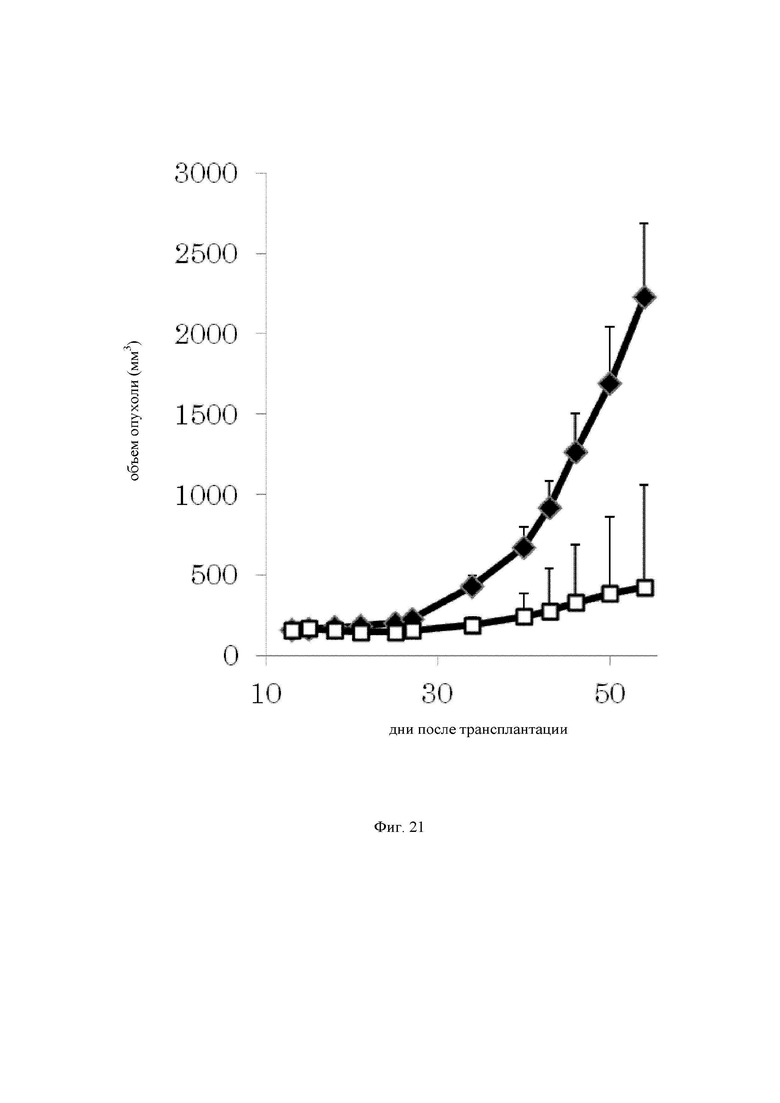
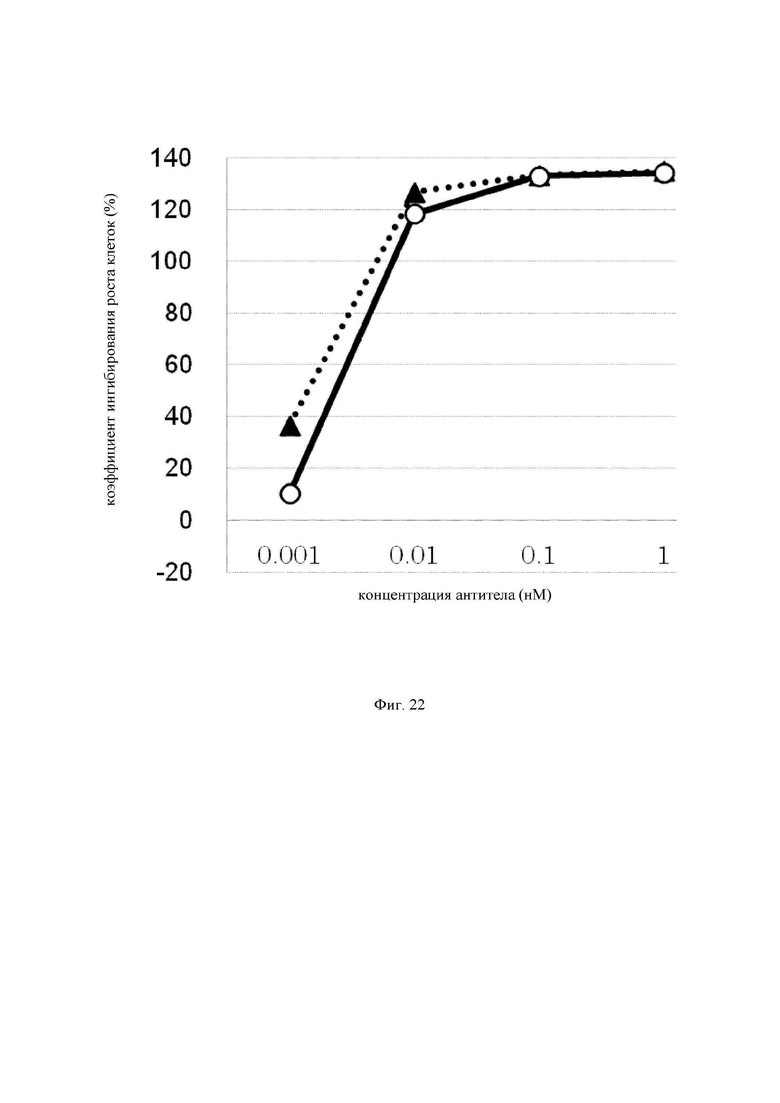
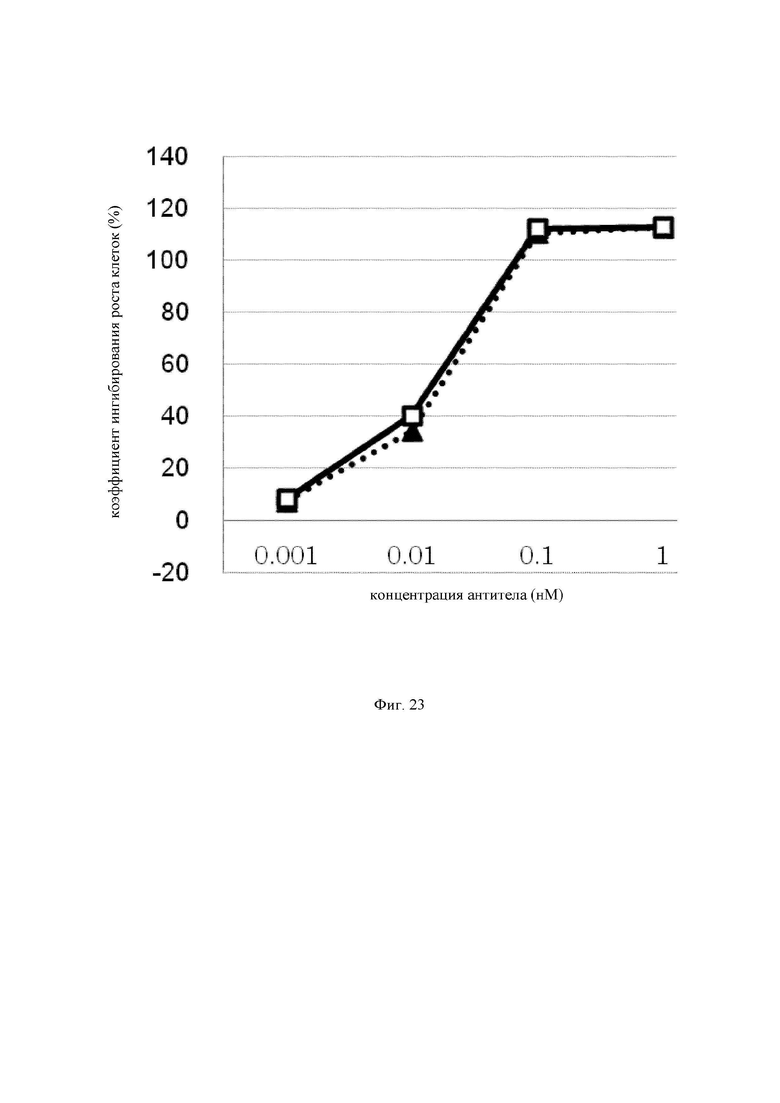
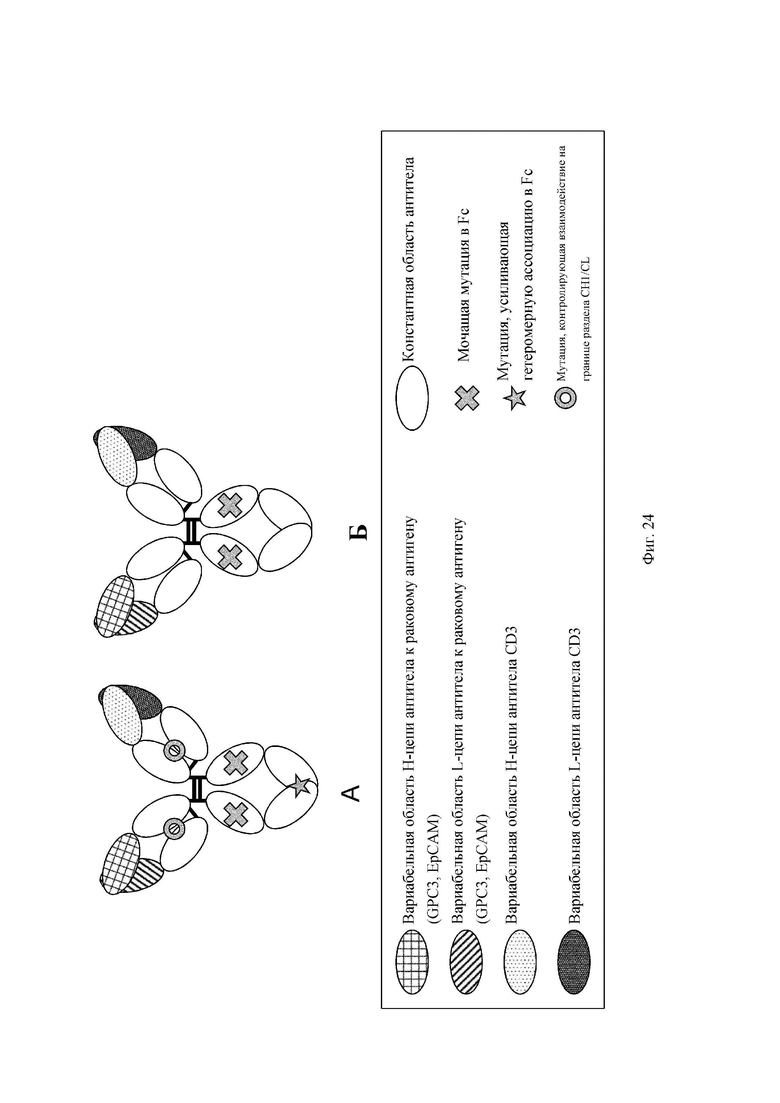
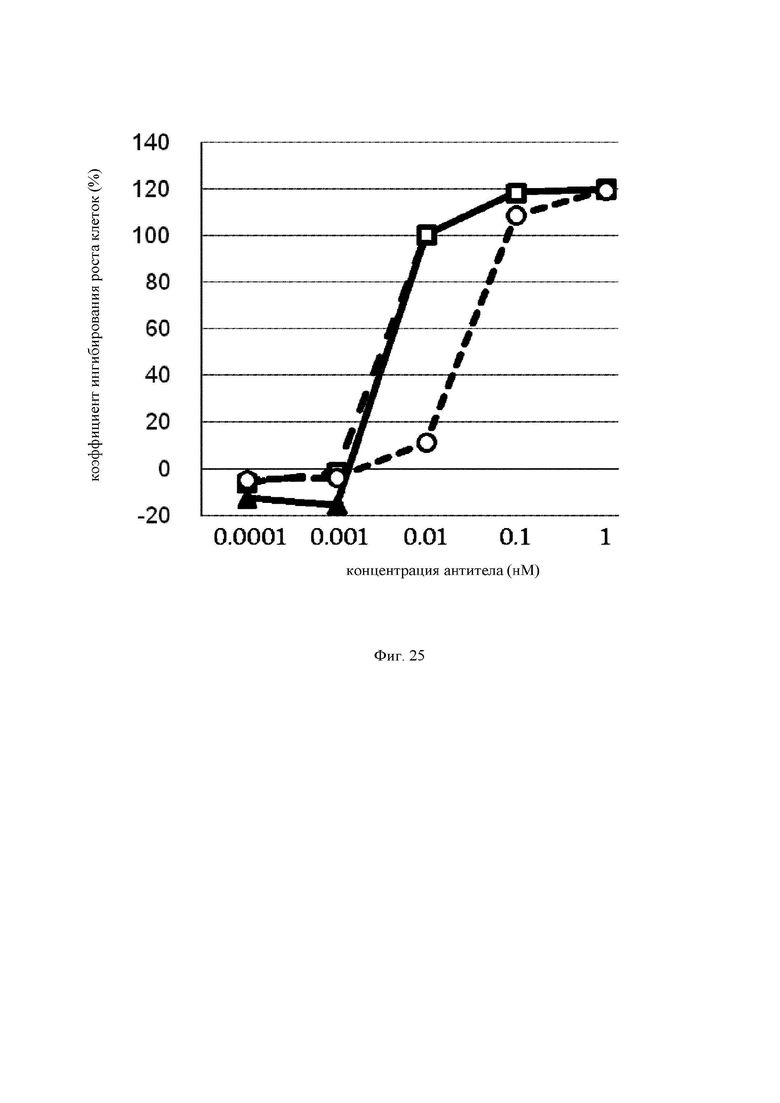
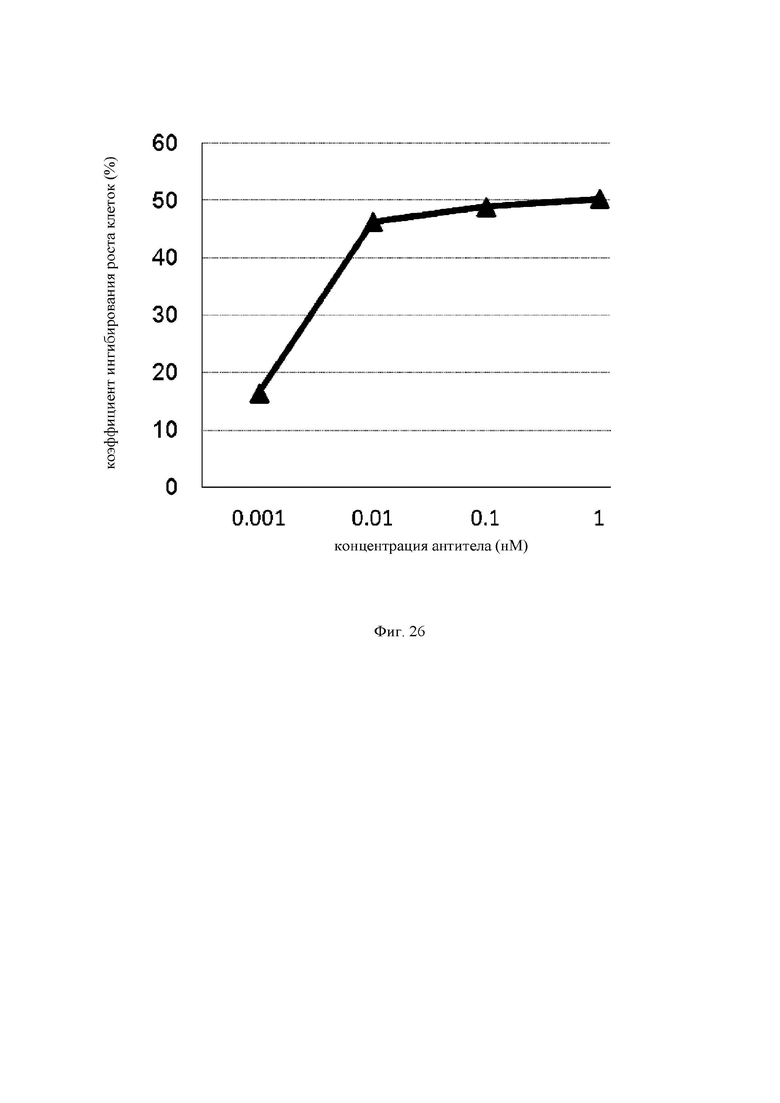
Изобретение относится к биотехнологии. Описано биспецифическое антитело, обладающее клеточной цитотоксичностью, которое включает: первую легкую цепь, содержащую первый домен VL и первый домен CL; вторую легкую цепь, содержащую второй домен VL и второй домен CL. При этом первая тяжелая цепь содержит первую вариабельную область тяжелой цепи и первую константную область тяжелой цепи. Первая константная область тяжелой цепи представляет собой константную область не дикого типа, содержащую одну из последовательностей SEQ ID NO: 23, 24, 25 и 26 с одной или более аминокислотными заменами. Вторая тяжелая цепь содержит вторую вариабельную область тяжелой цепи и вторую константную область тяжелой цепи. Причем вторая константная область тяжелой цепи является константной областью не дикого типа тяжелой цепи, содержащей одну из последовательностей SEQ ID NO: 23, 24, 25 и 26 с одной или более аминокислотными заменами. Аминокислотная последовательность второй константной области тяжелой цепи имеет тот же изотип, что и первая константная область тяжелой цепи. Первая легкая цепь и первая тяжелая цепь связываются с образованием первого антигенсвязывающего домена, который связывается с CD3. Вторая легкая цепь и вторая тяжелая цепь связываются с образованием второго антигенсвязывающего домена, который не связывается с CD3 и который связывается с раковым антигеном, который не является CD3. Первая константная область тяжелой цепи связывается со второй константной областью тяжелой цепи, где при измерении методом поверхностного плазмонного резонанса способность ассоциированных первой и второй константных областей тяжелой цепи связываться с данным человеческим рецептором Fсγ снижена по сравнению со способностью человеческого антитела IgG дикого типа того же изотипа, что и биспецифическое антитело, связываться с человеческим рецептором Fсγ в результате по меньшей мере одной из одной или более аминокислотных замен в пределах первой и второй константных областей тяжелой цепи. При этом по меньшей мере одна замена в первой и второй константных областях тяжелой цепи представляет собой изменение в аминокислотной последовательности по сравнению с аминокислотной последовательностью константных областей тяжелой цепи антитела IgG человека дикого типа и где по меньшей мере одна из аминокислот, которая приводит к пониженной способности связываться с человеческим рецептором Fсγ, находится в положении, выбранном из следующих положений согласно нумерации EU в каждой первой и второй константных областях тяжелой цепи: 220, 226, 229, 231, 232, 233, 234, 235, 236, 237, 238, 239, 240, 264, 265, 266, 267, 269, 270, 295, 296, 297, 298, 299, 300, 325, 327, 328, 329, 330, 331 и 332. Также раскрыта фармацевтическая композиция, содержащая указанное антитело и имеющая противораковую активность. Антитело по изобретению сохраняет сильную противораковую активность и имеет улучшенные характеристики безопасности, свойственные BiTE, имеет продолжительное время полужизни в крови и индуцирует цитотоксичность в отношении различных клеток. 2 н. и 46 з.п. ф-лы, 26 ил., 3 табл., 11 пр.
1. Биспецифическое антитело, обладающее клеточной цитотоксичностью, которое включает:
первую легкую цепь, содержащую первый домен VL и первый домен CL;
вторую легкую цепь, содержащую второй домен VL и второй домен CL;
первую тяжелую цепь, содержащую первую вариабельную область тяжелой цепи и первую константную область тяжелой цепи, причем первая константная область тяжелой цепи представляет собой константную область не дикого типа, содержащую одну из последовательностей SEQ ID NO: 23, 24, 25 и 26 с одной или более аминокислотными заменами и необязательно делецией аминокислот в положениях 446 и 447 согласно нумерации ЕU; и
вторую тяжелую цепь, содержащую вторую вариабельную область тяжелой цепи и вторую константную область тяжелой цепи, причем вторая константная область тяжелой цепи является константной областью не дикого типа тяжелой цепи, содержащей одну из последовательностей SEQ ID NO: 23, 24, 25 и 26 с одной или более аминокислотными заменами и необязательно делецией аминокислот в положениях 446 и 447 согласно нумерации EU, где аминокислотная последовательность второй константной области тяжелой цепи является такой же или отличается от аминокислотной последовательности первой константной области тяжелой цепи и имеет тот же изотип, что и первая константная область тяжелой цепи;
где первая легкая цепь и первая тяжелая цепь связываются с образованием первого антигенсвязывающего домена, который связывается с CD3, где вторая легкая цепь и вторая тяжелая цепь связываются с образованием второго антигенсвязывающего домена, который не связывается с CD3 и который связывается с раковым антигеном, который не является CD3, при этом первая константная область тяжелой цепи связывается со второй константной областью тяжелой цепи, где при измерении методом поверхностного плазмонного резонанса способность ассоциированных первой и второй константных областей тяжелой цепи связываться с данным человеческим рецептором Fсγ снижена по сравнению со способностью человеческого антитела IgG дикого типа того же изотипа, что и биспецифическое антитело, связываться с человеческим рецептором Fсγ, в результате по меньшей мере одной из одной или более аминокислотных замен в пределах первой и второй константных областей тяжелой цепи, где по меньшей мере одна замена в первой и второй константных областях тяжелой цепи представляет собой изменение в аминокислотной последовательности по сравнению с аминокислотной последовательностью константных областей тяжелой цепи антитела IgG человека дикого типа и где по меньшей мере одна из аминокислот, которая приводит к пониженной способности связываться с человеческим рецептором Fсγ, находится в положении, выбранном из следующих положений согласно нумерации EU в каждой первой и второй константных областях тяжелой цепи: 220, 226, 229, 231, 232, 233, 234, 235, 236, 237, 238, 239, 240, 264, 265, 266, 267, 269, 270, 295, 296, 297, 298, 299, 300, 325, 327, 328, 329, 330, 331 и 332.
2. Биспецифическое антитело по п. 1, где
аминокислотная последовательность в положениях с 118 по 260 (согласно нумерации EU) первой константной области тяжелой цепи и второй константной области тяжелой цепи представляет собой последовательность соответствующей части SEQ ID NO: 24; или аминокислотная последовательность в положениях с 261 по 447 (согласно нумерации EU) первой константной тяжелой цепи и второй константной области тяжелой цепи представляет собой последовательность соответствующей части SEQ ID NO: 26.
3. Биспецифическое антитело по п. 1, где первая константная область тяжелой цепи и вторая константная область тяжелой цепи каждая включает последовательность SEQ ID NO: 23 с одной или более заменами и, необязательно, делецию двух остатков на карбоксильном конце SEQ ID NO: 23.
4. Биспецифическое антитело по п. 3, где одна или более замен включают замену как в первой, так и во второй константных областях тяжелой цепи в положении, выбранном из положений 233, 234, 235, 236, 327, 330 и 331 (согласно нумерации EU), причем замена в выбранном положении является аминокислотой из соответствующего положения в IgG2 или IgG4.
5. Биспецифическое антитело по п. 3, где одна или более замен включают замену как в первой, так и во второй константных областях тяжелой цепи в положении 234, 235 или 297 (согласно нумерации ЕС).
6. Биспецифическое антитело по п. 5, где одна или более замен включают замену аланином.
7. Биспецифическое антитело по п. 1, где аминокислотная последовательность второй константной области тяжелой цепи отличается от аминокислотной последовательности первой константной области тяжелой цепи.
8. Биспецифическое антитело по п. 7, где:
в одной из двух константных областей тяжелой цепи биспецифического антитела аминокислоты в положениях 349 и 366 (согласно нумерации ЕU) представляют собой цистеин и триптофан соответственно;
и в другой константной области тяжелой цепи биспецифического антитела аминокислоты в положениях 356, 366, 368 и 407 (согласно нумерации ЕU) представляют собой цистеин, серин, аланин и валин соответственно.
9. Биспецифическое антитело по п. 7, где:
в одной из двух константных областей тяжелой цепи биспецифического антитела аминокислота в положении 356 представляет собой лизин, в другой константной области тяжелой цепи биспецифического антитела аминокислота в положении 439 представляет собой глутаминовую кислоту;
и в одной из двух константных областей тяжелой цепи биспецифического антитела, но не в обеих, аминокислота в положении 435 является аргинином (согласно нумерации ЕU).
10. Биспецифическое антитело по п. 8, где аминокислоты согласно нумерации ЕU в положениях 446 и 447 делетированы в обеих константных областях тяжелой цепи биспецифического антитела.
11. Биспецифическое антитело по п. 1, где человеческий рецептор Fсγ выбран из FcγI, FcγIIA, FcγIIB, FcγIIIA и FcγIIIB.
12. Биспецифическое антитело по п. 1, где аминокислотная последовательность второй константной области тяжелой цепи является такой же, как и аминокислотная последовательность первой константной области тяжелой цепи.
13. Биспецифическое антитело по п. 1, где аминокислотная последовательность второй константной области тяжелой цепи отличается от аминокислотной последовательности первой константной области тяжелой цепи и где процентное содержание гетеродимеров, образованных в смеси первой и второй тяжелых цепей, выше процентного содержания гетеродимеров, которые были бы связаны, если бы две константные области тяжелой цепи были идентичными и соответствовали одной из SEQ ID NO: 23-26.
14. Биспецифическое антитело по п. 13, где отличие между последовательностями первой и второй константных областей тяжелой цепи содержит отличие в одном или более положениях CH3 домена.
15. Биспецифическое антитело по п. 14, где отличие между последовательностями первой и второй константных областей тяжелой цепи дополнительно содержит отличие в одном или более положениях CH1 домена, при этом одно или более положение CH1 домена взаимодействует с CL доменом.
16. Биспецифическое антитело по п. 1, где аминокислотная последовательность второй константной области тяжелой цепи отличается от аминокислотной последовательности первой константной области тяжелой цепи и это отличие в последовательностях включает отличие в одном или более положениях CH1 домена двух константных областей тяжелой цепи, при этом одно или более положений CH1 домена взаимодействует с CL доменом.
17. Биспецифическое антитело по п. 1, где человеческий рецептор Fсγ выбран из FсγI, FcIIIγA и FcγIIIB.
18. Биспецифическое антитело по п. 1, где при измерении методом поверхностного плазмонного резонанса способность биспецифического антитела связываться с каждым из человеческих рецепторов Fсγ, FсγI, FcγIIIA и FcγIIIB снижена по сравнению со способностью человеческого IgG антитела дикого типа того же изотипа, такого как биспецифическое антитело, связываться с теми же человеческими рецепторами Fcγ.
19. Биспецифическое антитело по п. 1, где при измерении методом поверхностного плазмонного резонанса способность биспецифического антитела связываться с каждым из человеческих рецепторов Fсγ, FсγI, FcγIIA, FcγIIB, FcγIIIA и FcγIIIB снижается по сравнению со способностью человеческого антитела IgG дикого типа того же изотипа, такого как биспецифическое антитело, связываться с тем же человеческим рецептором Fсγ.
20. Биспецифическое антитело по п. 1, где константная область первой и второй тяжелой цепи является константной областью тяжелой цепи человека.
21. Биспецифическое антитело по п. 1, где ассоциированные первая и вторая константные области тяжелых цепей по существу не имеют обнаруживаемой способности связываться с данным человеческим рецептором Fсγ при измерении методом поверхностного плазмонного резонанса.
22. Биспецифическое антитело по п. 1, где ассоциированное первое и второе тяжелые константные области цепи по существу не имеют обнаруживаемой способности связываться с человеческими рецепторами Fсγ, FсγI, FcγIIA, FcγIIB, FcγIIIA и FcγIIIB при измерении методом поверхностного плазмонного резонанса.
23. Биспецифическое антитело по п. 3, где одна или более замен включают одну из следующих наборов замен как в первой, так и во второй константных областях тяжелой цепи (согласно нумерации ЕU):
(a) L234F, L235E, P331S,
(b) C226S, C229S, P238S;
(c) C226S, C229S;
(d) C226S, C229S, E233P, L234V, L235A.
24. Биспецифическое антитело по п. 1, где каждая первая и вторая константная область тяжелых цепей включает последовательность SEQ ID NO: 24 с одной или несколькими заменами и необязательно делецию двух остатков на карбоксильном конце SEQ ID NO: 24.
25. Биспецифическое антитело по п. 24, где одна или более замен включают один из следующих наборов замен как в первой, так и во второй константной области тяжелой цепи (согласно нумерации ЕU):
(a) H268Q, V309L, A330S и P331 S;
(b) V234A;
(c) G237A;
(d) V234A и G237A;
(e) A235E и G237A; или
(f) V234A, A235E и G237A.
26. Биспецифическое антитело по п. 1, где первая константная область тяжелой цепи и вторая константная область тяжелой цепи включают последовательность SEQ ID NO: 25 с одной или более заменами и необязательно делецию двух остатков на карбоксильном конце SEQ ID NO: 25.
27. Биспецифическое антитело по п. 26, где одна или более замен включают одну из следующих замен как в первой, так и во второй константной области тяжелой цепи (согласно нумерации ЕU):
(a) F241A;
(b) D265A; или
(c) V264A.
28. Биспецифическое антитело по п. 1, где первая константная область тяжелой цепи и вторая константная область тяжелой цепи содержат последовательность SEQ JD NO: 26 с одной или несколькими заменами и необязательно делецию двух остатков на карбоксильном конце SEQ ID NO: 26.
29. Биспецифическое антитело по п. 28, где одна или более замен включают один из следующих наборов замен как в первой, так и во второй константной области тяжелой цепи (согласно нумерации ЕU):
(а) L235A, G237A и E318A;
(b) L235E; или
(c) F234A и L235A.
30. Биспецифическое антитело по п. 1, где одна или более замен включают замену в положении 234 (согласно нумерации ЕU) как в первой, так и во второй константной области тяжелой цепи.
31. Биспецифическое антитело по п. 1, где одна или более замен включают замену в положении 235 (согласно нумерации ЕU) как в первой, так и во второй константной области тяжелой цепи.
32. Биспецифическое антитело по п. 1, где одна или более замен включают замену в положении 239 (согласно нумерации ЕU) как в первой, так и во второй константной области тяжелой цепи.
33. Биспецифическое антитело по п. 1, где одна или более замен включают замену в положении 297 (согласно нумерации ЕU) как в первой, так и во второй константной области тяжелой цепи.
34. Биспецифическое антитело по п. 1, где остатки в положениях 234 и 235 (согласно нумерации ЕU) являются аланином как в первой, так и во второй константных областях тяжелой цепи.
35. Биспецифическое антитело по п. 1, в котором остатки в положениях 234, 235 и 297 (согласно нумерации ЕU) все являются аланином как в первой, так и во второй константной области тяжелой цепи.
36. Биспецифическое антитело по п. 1, где положения 446 и 447 (согласно нумерации ЕU) удалены в последовательности каждой из первой и второй константных областей тяжелой цепи.
37. Биспецифическое антитело по п. 1, где единственная(ые) замена(ы) в аминокислотной последовательности одной или обеих из первой и второй константных областей тяжелой цепи по сравнению с аминокислотной последовательностью SEQ ID NO: 23 находятся в положении(ях), независимо выбранных из следующих положений согласно нумерации ЕU: 220, 226, 229, 231, 232, 233, 234, 235, 236, 237, 238, 239, 240, 264, 265, 266, 267, 269, 270, 295, 296, 297, 298, 299, 300, 325, 327, 328, 329, 330, 331 и 332.
38. Биспецифическое антитело по п. 1, где единственная(ые) замена(ы) в аминокислотной последовательности одной или обеих из первой и второй константных областей тяжелой цепи по сравнению с аминокислотной последовательностью SEQ ID NO: 24 находятся в положении(ях), независимо выбранных из следующих положений согласно нумерации ЕU: 220, 226, 229, 231, 232, 233, 234, 235, 236, 237, 238, 239, 240, 264, 265, 266, 267, 269, 270, 295, 296, 297, 298, 299, 300, 325, 327, 328, 329, 330, 331, и 332.
39. Биспецифическое антитело по п. 1, где единственная(ые) замена(ы) в аминокислотной последовательности одной или обеих из первой и второй константных областей тяжелой цепи по сравнению с аминокислотной последовательностью SEQ ID NO: 25 находятся в положении(ях), независимо выбранных из следующих положений согласно нумерации ЕU: 220, 226, 229, 231, 232, 233, 234, 235, 236, 237, 238, 239, 240, 264, 265, 266, 267, 269, 270, 295, 296, 297, 298, 299, 300, 325, 327, 328, 329, 330, 331 и 332.
40. Биспецифическое антитело по п. 1, где единственная(ые) замена(ы) в аминокислотной последовательности одной или обеих из первой и второй константных областей тяжелой цепи по сравнению с аминокислотной последовательностью SEQ ID NO: 26 находятся в положении(ях), независимо выбранных из следующих положений согласно нумерации ЕU: 220, 226, 229, 231, 232, 233, 234, 235, 236, 237, 238, 239, 240, 264, 265, 266, 267, 269, 270, 295, 296, 297, 298, 299, 300, 325, 327, 328, 329, 330, 331 и 332.
41. Биспецифическое антитело по п. 1, где аминокислотная последовательность одной или обеих первой и второй константной области тяжелой цепи имеет замену по сравнению с аминокислотной последовательностью SEQ ID NO: 23 в положении(ях), независимо выбранных из следующих положений согласно нумерации ЕU: 229, 239, 240, 264, 265, 267, 297, 298, 299, 329, 331 и 332.
42. Биспецифическое антитело по п. 1, где аминокислотная последовательность одной или обеих первой и второй константной области тяжелой цепи имеет замену по сравнению с аминокислотной последовательностью SEQ ID NO: 24 в положении(ях), независимо выбранных из следующих положений согласно нумерации ЕU: 229, 239, 240, 264, 265, 267, 297, 298, 299, 329, 331 и 332.
43. Биспецифическое антитело по п. 1, где аминокислотная последовательность одной или обеих первой и второй константной области тяжелой цепи имеют замену по сравнению с аминокислотной последовательностью SEQ ID NO: 25 в положении(ях), независимо выбранных из следующих положений согласно нумерации ЕU: 229, 239, 240, 264, 265, 267, 297, 298, 299, 329, 331 и 332.
44. Биспецифическое антитело по п. 1, где аминокислотная последовательность одной или обеих первой и второй константной области тяжелой цепи имеют замену по сравнению с аминокислотной последовательностью SEQ ID NO: 26 в положении(ях), независимо выбранных из следующих положений согласно нумерации ЕU: 229, 239, 240, 264, 265, 267, 297, 298, 299, 329 и 332.
45. Биспецифическое антитело по п. 1, где биспецифическое антитело является человеческим или гуманизированным антителом.
46. Биспецифическое антитело по п. 1, где изотипом биспецифического антитела является человеческий IgG1, человеческий IgG2, человеческий IgG3 или человеческий IgG4.
47. Биспецифическое антитело по п. 1, где изотип каждой первой и второй константной области тяжелой цепи независимо выбирают из человеческого IgG1, человеческого IgG2, человеческого IgG3 и человеческого IgG4.
48. Фармацевтическая композиция для лечения рака, где фармацевтическая композиция в качестве активного ингредиента содержит биспецифическое антитело по любому из пп. 1-47.
| FISCHER et al., "Bispecific Antibodies: Molecules That Enable Novel Therapeutic Strategies Pathobiology", 2007, 74(1), рр.3-14 | |||
| BOHUA LI et al., "Construction and characterization of a humanized anti-human CD3 monoclonal antibody 12F6 with effective immunoregulation function", Immunology | |||
| Способ обработки целлюлозных материалов, с целью тонкого измельчения или переведения в коллоидальный раствор | 1923 |
|
SU2005A1 |
| MCEARCHEM et al., | |||
Авторы
Даты
2024-08-07—Публикация
2022-04-15—Подача