Изобретение относится к области биотехнологии и биомедицины, а именно к олигонуклеотидным аптамерам, способным специфично и с высоким сродством узнавать белок Dickkopf-1 (DKK-1) человека.
Аптамеры - это синтетические молекулы ДНК и РНК, которые специфично и с высоким сродством связывают определенные молекулярные мишени за счет сочетания уникальной нуклеотидной последовательности и сложной пространственной структуры. Олигонуклеотидные аптамеры, так же как пептиды, полученные методом фагового дисплея, или моноклональные антитела, («мАт») способны связываться с выбранными мишенями и модулировать их активность, либо обеспечивать специфическое узнавание мишеней.
По аффинности и специфичности связывания мишеней аптамеры сравнимы с моноклональными антителами, при этом обладают целым рядом уникальных преимуществ, таких как стабильность в более широком диапазоне условий, способность выдерживать циклы денатурации/ренатурации без потери сродства к мишени, возможность воспроизводимого, масштабируемого, автоматического химического синтеза, минимальная вариабельность основных характеристик от партии к партии [1].
Аптамеры находят широкое применение в качестве биоспецифичных элементов при создании тест-систем для детекции определенных молекул-аналитов, на данный момент доступно широкое разнообразие способов конструирования и методов получения аналитического сигнала в таких системах [2]. Особое внимание привлекают аптамерные тест-системы для детекции биомаркеров различных заболеваний.
Ближайшими функциональными аналогами аптамеров, широко используемыми сейчас в тест-системах для клинической лабораторной диагностики, являются антитела. Недостатками систем на основе антител являются высокая стоимость, а также недостаточная стабильность характеристик, связанная с изменчивостью свойств антител к одному и тому же антигену от производителя к производителю и от партии к партии у одного производителя [3]. Проблема стабильности характеристик становится особенно важной при проведении долговременных многолетних исследований, при которых необходим регулярный мониторинг уровней одних и тех же биомаркеров [4]. Преимущества аптамеров позволяют рассматривать их в качестве альтернативы антителам в диагностических системах.
На данный момент предложено большое количество различных тест-систем на основе аптамеров для детекции биомаркеров инфекционных, сердечно-сосудистых, онкологических заболеваний [5-7]. Однако большинство из них пока опробовано лишь на уровне принципиальной работоспособности (proof of principle), хотя несколько аптамерных тест-систем доступны в качестве коммерческих наборов [8].
Спондилоартриты (СпА) - группа хронических воспалительных ревматических заболеваний с преимущественным поражением позвоночника и крестцово-подвздошных суставов, включающая аксиальный спондилоартрит, псориатический артрит, реактивный артрит, артрит, связанный с хроническим воспалительным заболеванием кишечника, и недифференцированный спондилоартрит. Распространенность СпА варьируется от 0.5% до 2%, а пик заболеваемости приходится на молодой трудоспособный возраст. Так же для СпА характерен высокий уровень инвалидизации, снизить который представляется возможным только при условии своевременной диагностики заболевания и его осложнений, адекватной терапевтической тактики [9]. При этом на сегодняшний день постановка диагноза СпА представляет определенную трудность: от первых симптомов до подтверждения диагноза проходит в среднем 5.7 лет [10], что связано в том числе с несовершенством существующих диагностических подходов и классификационных критериев [11].
Современные направления терапии СпА носят преимущественно патогенетический характер: снижение воспаления и купирование процессов патологического ремоделирования костной ткани. Показано, что при развитии СпА патологическое ремоделирование костной ткани может проявляться как в виде потери костной массы, так и в форме остеопролиферации с образованием синдесмофитов. Эти процессы могут протекать параллельно у одного пациента, приводить к формированию полного анкилоза суставов и позвоночника, к низкоэнергетическим переломам на фоне вторичного остеопороза, что обуславливает высокий уровень стойкой утраты трудоспособности пациентов [12].
Таким образом, поиск и определение биомаркеров костного ремоделирования при СпА является актуальной для клинической практики проблемой, решение которой позволит дополнить существующие диагностические и терапевтические подходы для более быстрой и точной верификации диагноза, определения прогрессирования заболевания и подбора методов лечебного воздействия.
Биомаркеры и методы их оценки при патологическом ремоделировании костной ткани на сегодняшний день в полной мере не разработаны. В регулировании этого процесса принимают участие ряд белков, наибольший интерес из которых при СпА вызывает белок Dickkopf-1 (Dkk-1) - антагонист Wnt-сигнального пути [13-15]. Препятствием на пути систематических исследований роли данного маркера при спондилоартритах является отсутствие общепринятого стандартного метода для определения уровня Dkk-1 в сыворотке крови.
Известен способ количественного определения уровней Dkk-1 в сыворотке крови путем иммуноферментного анализа [16; 17]. К недостаткам данного способа относятся достаточно высокая стоимость анализа и проблемы с воспроизводимостью результатов, полученных с использованием разных партий антител и диагностических наборов, что особенно значимо для длительных многолетних исследований. Именно поэтому более выгодной альтернативой антителам могут стать аптамеры к белку DKK-1.
Наиболее близким к заявляемому ДНК-аптамеру - прототипом, является аптамер TD10 к белку DKK-1 человека, представляющий собой 39-звенный ДНК-аптамер, имеющий молекулярную массу 12.1 кДа, с константой диссоциации 7.7 нМ [18]. Данный аптамер обладает специфическим сродством к белку-мишени и был опробован в качестве компонента тест-систем для детекции DKK-1 в крови больных гепатоцеллюлярной карциномой.
Задачей настоящего изобретения является получение новых ДНК-аптамеров, обладающих способностью специфично и прочно связывать белок DKK-1 человека,
Технический результат: повышение аффинности ДНК-аптамеров, специфично и прочно связывающихся с белком Dickkopf-1 человека, а также расширение ассортимента ДНК-аптамеров.
Поставленная задача достигается созданием ДНК-аптамеров DK1, DK2, DK4, DK4-4H, DK2-48t, представляющих собой олигодезоксирибонуклеотиды с числом нуклеотидных звеньев от 41 до 71, имеющих нуклеотидные последовательности: SEQ ID NO 1, SEQ ID NO 2, SEQ ID NO 3, SEQ ID NO 4, SEQ ID NO 5 соответственно, представленные в перечне последовательностей.
Нуклеотидные последовательности 71-звенных аптамеров DK1, DK2, DK4 установлены с помощью метода селекции in vitro из комбинаторной 71-звенной библиотеки ДНК с использованием в качестве мишени рекомбинантного белка DKK-1 человека. После установления нуклеотидных последовательностей индивидуальных аптамеров проводили скрининг аффинности и специфичности их связывания с белком-мишенью. В результате минимизации нуклеотидной последовательности (удаление из нуклеотидной последовательности фрагментов, не участвующих в связывании с мишенью) были получены 48-звенный аптамер DK2-48t и 41-звенный аптамер DK4-4U.
Нуклеотидные последовательности ДНК-аптамеров DK1(SEQ ID NO 1), DK2(SEQ ID NO 2), DK4(SEQ ID NO 3), DK4-4U (SEQ ID NO 4), DK2-48t (SEQ ID NO 5) представлены в перечне последовательностей.
Полученные аптамеры обладали следующими характеристиками:
- формировали прочные комплексы с рекомбинантным белком DKK-1 человека;
- не связывались с мажорным белком сыворотки крови человека - человеческим сывороточным альбумином.
Синтез заявляемого ДНК-аптамера по изобретению осуществляют на автоматическом ДНК/РНК-синтезаторе ASM-800 с использованием стандартного твердофазного фосфитамидного метода.
Определяющими отличиями предлагаемых ДНК-аптамеров от прототипа являются:
- уникальные нуклеотидные последовательности, негомологичные нуклеотидной последовательности прототипа, аптамера TD10;
- более высокая аффинность к белку-мишени по сравнению с прототипом TD10 при анализе аффинности в одинаковых условиях (оценена по значениям констант диссоциации комплекса аптамер-мишень).
Изобретение иллюстрируется следующими примерами.
Пример 1. Получение аптамеров DK1, DK2, DK4, DK4-41t, DK2-48t
Синтез ДНК-аптамеров DK1, DK2, DK4, DK4-41t, DK2-48t проводили на автоматическом ДНК/РНК синтезаторе ASM-800 (Биоссет, Россия) по оптимизированным для данного прибора протоколам [19] с использованием β-цианэтил-N,N-диизопропилфосфитамидов 5',N-защищенных 2'-дезоксирибонуклеозидов. В качестве полимерного носителя использовали стеклянные частицы CPG (controlled pore glass) с диаметром пор 500 А с присоединенным через 3'-гидроксильную группу 5', N-защищенным 2'-дезоксирибонуклеозидом.
Цикл синтеза включал:
1. Обработку 3%-ным раствором дихлоруксусной кислоты в дихлорметане для удаления защитной диметокситритильной группы с 5'-гидроксильной группы растущей цепи (стадия детритилирования);
2. Конденсацию следующего мономерного звена - β-цианэтил-N,N-бис-диизопропиламидофосфита защищенного дезоксирибонуклеозида в концентрации 0,1 М в ацетонитриле. В качестве активирующего агента на данной стадии использовали 5-этилтио-1Н-тетразол в концентрации 0,25 М в ацетонитриле (стадия конценсации);
3. Блокирование непрореагировавших 5'-гидроксильных групп с использованием смесей пропионовый ангидрид/2,6-лутидин/тетрагидрофуран (10:10:80, v/v/v) и N-метилимидазол/тетрагидрофуран (16:84, v/v) (стадия кэпирования);
4. Превращение фосфиттриэфирных межнуклеозидных связей в фосфотриэфирные обработкой 0.02 М раствором йода в смеси пиридин/вода/тетрагидрофуран (1:9:90, v/v/v) (стадия окисления).
После проведения необходимого числа циклов синтеза и удаления 5'-концевой диметокситритильной группы получали защищенный полимерсвязанный ДНК-аптамер.
Для отделения олигонуклеотидов от полимерного носителя, деблокирования гетероциклических оснований и межнуклеотидных фосфатов к защищенному полимерсвязанному аптамеру добавляли 300 мкл смеси АМА (40%-ный водный раствор метиламина/28%-ный водный раствор аммиака, 1:1, v/v) и выдерживали в течение 15 мин при 65°С и постоянном перемешивании. После охлаждения при -20°С в течение 15 мин раствор отделяли от полимерного носителя центрифугированием. Полимер промывали 2×150 мкл смеси этанол : ацетонитрил : вода (1:1:1, v/v/v). Все растворы объединяли и упаривали до объема 100 мкл в вакуумном испарителе Concentrator Plus (Eppendorf).
Полностью деблокированные аптамеры выделяли препаративным электрофорезом в денатурирующем 12 или 15%-ном акриламидном геле (акриламид : N,N'-метиленбисакриламид (30:1), 8 М мочевина, 50 мМ Трис-борат (рН 8.3), 0.1 мМ Na2EDTA) при напряжении 50 В/см. После проведения электрофореза олигонуклеотид визуализировали в УФ-свете, затем вырезали участки геля, содержащие олигонуклеотиный материал, и элюировали 0.3 М водным раствором NaClO4. Элюат обессоливали с помощью картриджа Waters SepPac С18, упаривали до объема 100 мкл и осаждали аптамер добавлением 10-ти кратного избытка 2%-ного раствора NaClO4 в ацетоне, и центрифугировали в течение 5 мин при 13200 об./мин. Супернатант отбирали, осадок олигонуклеотида промывали ацетоном, высушивали при комнатной температуре.
ДНК-аптамеры анализировали методом аналитического гель-электрофореза в 15%-ном денатурирующем ПААГ с последующим окрашиванием геля 0,1%-ным раствором красителя "Stains-All" в смеси формамид/вода (1:1, v/v).
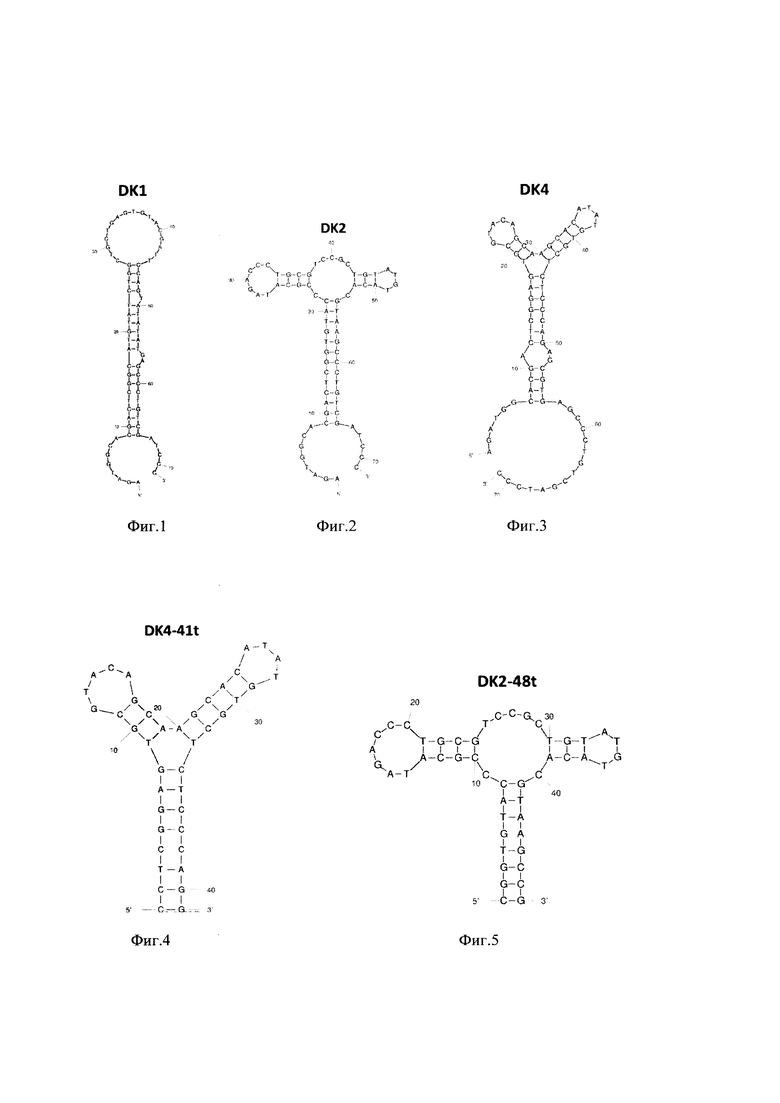
На фиг. 1-5 представлены вторичные структуры ДНК-аптамеров DK1, DK2, DK4, DK4-4U, DK2-48t по данным компьютерного моделирования, проведенного на основе алгоритма минимизации свободной энергии с помощью программного пакета mfold version 2.3 [20].
Пример 2. Определение сродства аптамеров DK1, DK2, DK4, DK4-4U, DK2-48t к белку DKK-1
Для определения сродства аптамеров DK1, DK2, DK4, DK4-41t, DK2-48t к белку DKK-1 использовали гетерофазный колориметрический метод в микропланшетном варианте. В лунки планшета с высокой сорбционной емкостью (Greiner) вносили по 100 мкл раствора DKK-1 с концентрацией 2 мкг/мл в фосфатно-солевом буфере (PBS), рН 7.4. Инкубировали при 4°С в течение 16 ч, раствор удаляли, лунки промывали 3 раза буфером для промывки (PBS, рН 7.4, 5 мМ MgCl2, 0.05% Твин-20) по 300 мкл. Для блокирования свободной поверхности лунок вносили по 150 мкл 1% бычьего сывороточного альбумина в PBS и инкубировали при 25°С в течение 2 ч при постоянном перемешивании, затем удаляли раствор и промывали 3 раза по 300 мкл буфера для промывки. Конъюгаты аптамеров с биотином (0-200 нМ) в PBS, рН 7.4, 5 мМ MgCb, 0.05% Твин-20 инкубировали 5 мин при 95°С, 5 мин выдерживали во льду и не менее 15 мин при 25°С, после чего вносили по 100 мкл в лунки планшета и инкубировали 1 ч при 25°С и постоянном перемешивании. Раствор удаляли и промывали лунки 3 раза по 300 мкл буфера для промыки. Добавляли по 100 мкл 0.2 мкл/мл конъюгата стрептавидина с пероксидазой хрена, инкубировали при 25°С и постоянном перемешивании 1 ч. Раствор удаляли, лунки промывали 3 раза по 300 мкл буфера для промывки. Для получения сигнала вносили по 100 мкл раствора 3,3',5,5'-тетраметилбензидина с концентрацией 0.2 мг/мл в 0.1 М натрий-ацетатном буфере (рН 5.2), содержащего 0.03% Н2О2, инкубировали 20 мин при 25°С и постоянном перемешивании. Для фиксации окраски добавляли 100 мкл 10% H2SO4, затем измеряли оптическую плотность на длине волны 450 нм с использованием микропланшетного спектрофотометра AMR-100 (All-Sheng).
На фиг. 6 приведены кривые связывания с белком DKK-1 для ДНК-аптамеров DK1, DK2, DK4, DK4-41t, DK2-48t. Полученные данные свидетельствуют о том, что аптамеры DK1, DK2, DK4, DK4-4U, DK2-48t связываются с белком DKK-1 с высоким сродством. Рассчитанные из полученных данных значения константы диссоциации комплекса с белком для ДНК-аптамеров DK1, DK2, DK4, DK4-41t, DK2-48t составили 1.3±1 нМ, 1.8±1 нМ, 1.8±0.5 нМ, 5.6±3 нМ, 3.3±2 нМ, соответственно. Прототип - ДНК-аптамер TD10 в аналогичных условиях характеризуется значением константы диссоциации комплекса с белком DKK-1 7,7±4 нМ. Более низкие значения константы диссоциации для полученных нами аптамеров DK1, DK2, DK4, DK4-41t, DK2-48t указывают на образование более прочных комплексов и повышенное сродство к белку-мишени DKK-1 по сравнению с прототипом.
Пример 3. Использование ДНК-аптамера DK4 для детекции белка DKK-1 в сыворотке крови.
Проводили исследование возможности количественной детекции белка DKK-1 в сыворотках крови пациентов со спондилоартритами на примере образцов от пациентов с анкилозирующим спондилитом на ранней и развернутой стадии заболевания. Забор крови и все инструментальные исследования проводились после подписания информированного добровольного согласия. Кровь для исследования собирали в стандартные вакутейнеры объемом 5 мл с разделительным гелем и активатором свертывания. Образцы центрифугировали в течение 10 мин при 3000 об/мин, полученные сыворотки аликвотировали по 500 мкл и хранили при -20°С.
Использовали микропланшетный колориметрический формат анализа с улавливанием анализируемого белка при помощи ковалентно иммобилизованного в лунках планшета ДНК-аптамера. Для каждого образца, в том числе при построении калибровочной кривой, проводили два параллельных измерения.
Для иммобилизации аптамера в лунки коммерческого карбокси-модифицированного планшета Nunc NucleoLink добавляли по 100 мкл 250 нМ водного раствора аптамера DK4, содержащего 10 мМ 1-метилимидазола и 10 мМ 1-этил-3-(3-диметиламинопропил)карбодиимида. Инкубировали при 50°С и постоянном перемешивании в течение 16 ч. Раствор удаляли, лунки промывали 3 раза по 300 мкл буфера для промывки (PBS, рН 7.4, 5 мМ MgCl2, 0.05% Твин-20). Для блокирования свободной поверхности в лунки добавляли по 250 мкл 1% раствора бычьего сывороточного альбумина в 1x PBS и инкубировали при 25°С в течение 1 ч при постоянном перемешивании, затем раствор удаляли и промывали лунки 3 раза по 300 мкл буфера для промывки.
Для построения калибровочной кривой в лунки добавляли по 100 мкл 0-10 нг/мл раствора DKK-1 в 1x PBS, рН 7.4, 5 мМ MgCl2, 0.05% Твин-20.
Для проведения анализа образцы сыворотки предварительно разбавляли в 10 раз буфером PBS и вносили по 100 мкл образца в соответствующие лунки. Инкубировали 1 ч при перемешивании при 25°С. Затем растворы удаляли, лунки промывали 4 раза по 300 мкл буфера для промывки. Для получения колориметрического сигнала вносили по 100 мкл раствора конъюгата стрептавидина с пероксидазой хрена с концентрацией 0.2 мкл/мл, инкубировали при 25°С и постоянном перемешивании в течение 1 ч. Затем раствор удаляли, лунки промывали 5 раз по 300 мкл буфера для промывки. Вносили по 100 мкл раствор 0.2 мг/мл 3,3',5,5'-тетраметилбензидина в 0.1 М натрий-ацетатном буфере (рН 5.2), содержащий 0.03% Н2О2, инкубировали 20 мин при 25°С и постоянном перемешивании. Для фиксации окраски добавляли 100 мкл 10% H2SO4, затем измеряли оптическую плотность на 450 нм с использованием микропланшетного спектрофотометра AMR-100 (All-Sheng).
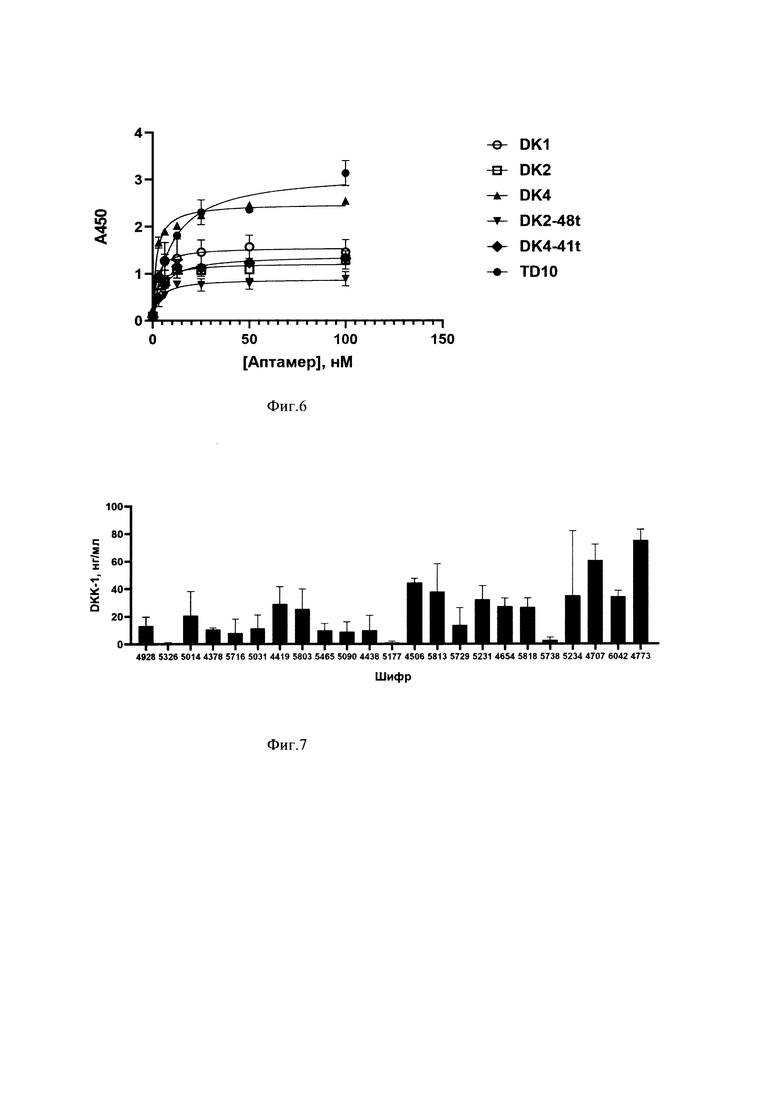
Для построения калибровочной кривой и расчета концентраций DKK-1 в образцах сыворотки использовали нелинейную аппроксимацию четырех-параметрическим логарифмическим уравнением. На фиг. 7 приведены усредненные по двум параллельным измерениям значения уровня DKK-1 в каждом из исследованных образцов сыворотки. Из полученных значений видно, что использование иммобилизованного в лунках планшета аптамера DK4 позволяет количественно определить уровни DKK-1 в образцах сыворотки крови от пациентов с анкилозирующим спондилитом колориметрическим методом. Полученные значения лежат в типичном диапазоне для содержания данного белка в сыворотке крови человека, что говорит о принципиальной возможности использования аптамера DK4 для колориметрического микропланшетного анализа сывороток крови от пациентов со спондилоартритами.
Использование предлагаемых ДНК-аптамеров позволит расширить ассортимент олигонуклеотидных аптамеров, способных высокоаффинно и специфично узнавать белок DKK-1 человека и пригодных для использования в качестве биоспецифичных элементов для создания тест-систем для детекции белка DKK-1 в крови при спондилоартритах.
Источники информации
1. Byun J. Recent Progress and Opportunities for Nucleic Acid Aptamers. Life. 2021 V. 11, P. 193. doi: 10.3390/life11030193.
2. Arshavsky-Graham S., Heuer C, Jiang X., Segal E. Aptasensors versus immunosensors-Which will prevail? Eng Life Sci. 2022, V. 22, P. 319-333. doi: 10.1002/elsc.202100148.
3. Bradbury A., Plückthun A. Reproducibility: Standardize antibodies used in research. Nature. 2015, V. 518, P. 27-29. doi: 10.1038/518027a.
4. Weller M. G. Quality Issues of Research Antibodies. Analytical Chemistry Insights. 2016, V. 11, P. 21-27. doi: 10.4137/ACI.S31614.
5. Rhouati A., Rhouati A., Marty J.L. A Review on Aptamers Selection and Application in Heart Diseases Diagnosis. 2022, V. 22, P. 2463-2473. doi: 10.2174/1568026622666220831114322.
6. Hu X., Zhang D., Zeng Z., Huang L., Lin X., Hong S. Aptamer-Based Probes for Cancer Diagnostics and Treatment. Life 2022, V. 12, P. 1937. doi: 10.3390/life12111937.
7. Chen J., Zhou J., Peng Y., Xie Y., Xiao Y. Aptamers: A prospective tool for infectious diseases diagnosis. J Clin Lab Anal. 2022, V. 36, P. e24725. doi: 10.1002/jcla.24725
8. Kaur H., Bruno J.G., Kumar A., Sharma Т.K. Aptamers in the Therapeutics and Diagnostics Pipelines. Theranostics. 2018, V 8, P. 4016-4032. doi: 10.7150/thno.25958.
9. Davydova A., Kurochkina Y., Goncharova V., Vorobyeva M., Korolev M. The interleukine-17 cytokine family: role in development and progression of spondyloarthritis, current and potential therapeutic inhibitors. Biomedicines. 2023, V. 11, P. 1328. doi: 10.3390/biomedicines11051328.
10. Redeker, I.; Callhoff, J.; Hoffmann, F.; Haibel, H.; Sieper, J.; Zink, A.; Poddubnyy, D. Determinants of diagnostic delay in axial spondyloarthritis: An analysis based on linked claims and patient-reported survey data. Rheumatology. 2019, V. 58, P. 1634-1638. doi: 10.1093/rheumatology/kez090.
11. Poddubnyy D. Classification vs diagnostic criteria: the challenge of diagnosing axial spondyloarthritis. Rheumatology (Oxford). 2020, V. 59, P. iv6-iv17. doi: 10.1093/rheumatology/keaa250.
12. Clunie G, Horwood N. Loss and gain of bone in spondyloarthritis: what drives these opposing clinical features? Therapeutic Advances in Musculoskeletal Disease. 2020, V. 12, P. 1759720X20969260. doi: 10.1177/1759720X20969260.
13. Daoussis D, Kanellou A, Panagiotopoulos E, Papachristou D. DKK-1 Is Underexpressed in Mesenchymal Stem Cells from Patients with Ankylosing Spondylitis and Further Downregulated by IL-17. International Journal of Molecular Sciences. 2022, V. 23, P. 6660. doi: 10.3390/ijms23126660.
14. Daoussis D., Andonopoulos A. P. The emerging role of Dickkopf-1 in bone biology: is it the main switch controlling bone and joint remodeling? Seminars in Arthritis and Rheumatism. 2011, V. 41, P. 170-177. doi: 10.1016/j.semarthrit.2011.01.006.
15. Nocturne G., Pavy S., Boudaud S., Seror R., Goupille P., Chanson P., van der Heijde D., van Gaalen F., Berenbaum F., Mariette X., Briot K., Feydy A., Claudepierre P., Dieude P., Nithitham J., Taylor K.E., Criswell L.A., Dougados M., Roux C, Miceli-Richard C. Increase in Dickkopf-1 serum level in recent spondyloarthritis. data from the DESIR cohort. PLOS One. 2015, V. 10, P. e0134974. doi: 10.1371/journal.pone.0134974.
16. Seror R., Boudaoud S., Pavy S., Nocturne G., Schaeverbeke Т., Saraux A., Chanson P., Gottenberg J.-E., Devauchelle-Pensec V., Tobon G.J., Mariette X., Miceli-Richard C. Increased Dickkopf-1 in recent-onset rheumatoid arthritis is a new biomarker of structural severity. Data from the ESPOIR cohort. Scientific Reports. 2016, V. 6, P. 18421. doi: 10.1038/srep 18421.
17. Y. Yuliasih. The increasing level of DKK-1 as a new bone formation factor in patients with early spondyloarthritis. Autoimmune Diseases. 2023, V. 2023, P. 5543234. doi: 10.1155/2023/5543234.
18. Zhou Y., Li W., Tseng Y., Zhang J., Liu J. Developing slow-off dickkopf-1 aptamers for early-diagnosis of hepatocellular carcinoma. Talanta. 2019, V. 194, P. 422-429. doi: 10.1016/j.talanta.2018.10.014.
19. Bellon L. Oligoribonucleotides with 2'-O-(tert-butyldimethylsilyl) groups. Current protocols in nucleic acids chemistry. 2001, S. 1, P. 3.6.1-3.6.13. doi: 10.1002/0471142700.nc0306s01.
20. Zuker M. Mfold web server for nucleic acid folding and hybridization prediction. Nucleic Acids Res. 2003, V. 31, P. 3406-3415. doi: 10.1093/nar/gkg595.
--->
Перечень последовательностей
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing SYSTEM "ST26SequenceListing_V1_3.dtd"
PUBLIC "-//WIPO//DTD Sequence Listing 1.3//EN">
-<ST26SequenceListing productionDate="2023-07-14"
softwareVersion="2.2.0" softwareName="WIPO Sequence"
fileName="aptDKK1.xml" dtdVersion="V1_3"
originalFreeTextLanguageCode="ru">
-<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>1</ApplicationNumberText>
<FilingDate>2023-07-14</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>1</ApplicantFileReference>
-<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>1</ApplicationNumberText>
<FilingDate>2023-07-14</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">Федеральное государственное
бюджетное учреждение науки Институт химической биологии и
фундаментальной медицины Сибирского отделения Российской академии
наук (ИХБФМ СО РАН)</ApplicantName>
<ApplicantNameLatin>The Institute of Chemical Biology and Fundamental
Medicine of the Siberian Branch of the Russian Academy of
Sciences</ApplicantNameLatin>
<InventionTitle languageCode="ru"> ДНК-аптамер, специфично
связывающийся с белком Dickkopf-1 человека </InventionTitle>
<SequenceTotalQuantity>5</SequenceTotalQuantity>
-<SequenceData sequenceIDNumber="1">
-<INSDSeq>
<INSDSeq_length>71</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
-<INSDSeq_feature-table>
-<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..71</INSDFeature_location>
-<INSDFeature_quals>
-<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
-<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agatggcacgactcggcatgtatctggctgctgagtgtacgattccagtata
tatgagccctgtcgatccc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
-<SequenceData sequenceIDNumber="2">
-<INSDSeq>
<INSDSeq_length>71</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
-<INSDSeq_feature-table>
-<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..71</INSDFeature_location>
-<INSDFeature_quals>
-<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
-<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agatggcacgactcggtgtacccgcatagaccctgcgtccgctgtatgtaca
cgtaagccctgtcgatccc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
-<SequenceData sequenceIDNumber="3">
-<INSDSeq>
<INSDSeq_length>71</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
-<INSDSeq_feature-table>
-<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..71</INSDFeature_location>
-<INSDFeature_quals>
-<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
-<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agatggcacgactcggagtgcgtacagcaagcacatatgtgctctcccagag
cgtgagccctgtcgatccc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
-<SequenceData sequenceIDNumber="4">
-<INSDSeq>
<INSDSeq_length>41</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
-<INSDSeq_feature-table>
-<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..41</INSDFeature_location>
-<INSDFeature_quals>
-<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
-<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cctcggagtgcgtacagcaagcacatatgtgctctcccagg</INSDSeq_s
equence>
</INSDSeq>
</SequenceData>
-<SequenceData sequenceIDNumber="5">
-<INSDSeq>
<INSDSeq_length>48</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
-<INSDSeq_feature-table>
-<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..48</INSDFeature_location>
-<INSDFeature_quals>
-<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
-<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cggtgtacccgcatagaccctgcgtccgctgtatgtacacgtaagccg</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| ДНК аптамеры, связывающие сердечный тропонин I человека | 2019 |
|
RU2716409C1 |
| РНК-аптамер, обладающий способностью узнавать аутоантитела, характерные для рассеянного склероза | 2017 |
|
RU2644229C1 |
| Способ получения конъюгатов олигонуклеотидов с кластерами бора | 2022 |
|
RU2786533C1 |
| Модифицированная направляющая РНК, обладающая способностью инактивировать систему редактирования генома CRISPR/Cas9, и способ ее получения | 2020 |
|
RU2765159C1 |
| Способ выявления опухолеспецифичных мишеней в гистологических срезах тканей больных раком легкого человека | 2015 |
|
RU2639238C2 |
| Рекомбинантная плазмидная ДНК pET19b-SAV, обеспечивающая синтез полноразмерного белка стрептавидина Streptomyces avidinii, штамм бактерий Escherichia coli - продуцент растворимого полноразмерного белка стрептавидина Streptomyces avidinii | 2019 |
|
RU2728652C1 |
| Однодоменное антитело ламы Н5 и его производное H5-Fc, специфически связывающие RBD-домен S-белка вируса SARS-CoV-2, обладающие вируснейтрализующей активностью | 2022 |
|
RU2793967C1 |
| Способ выявления мишеней, ассоциированных с определенным диагнозом, в крови пациентов на основе ДНК-аптамеров | 2017 |
|
RU2685936C2 |
| РНК-АПТАМЕР, ОБЛАДАЮЩИЙ СПОСОБНОСТЬЮ УЗНАВАТЬ ХАРАКТЕРНЫЕ ДЛЯ РАССЕЯННОГО СКЛЕРОЗА АУТОАНТИТЕЛА | 2014 |
|
RU2549704C1 |
| ДНК-маркер для селекции сортов ярового ячменя, устойчивых к темно-бурой пятнистости | 2020 |
|
RU2740404C1 |
Изобретение относится к области биотехнологии и медицины. Предложены новые ДНК-аптамеры: DK1, DK2, DK4, DK4-41t, DK2-48t, представляющие собой олигодезоксирибонуклеотиды с числом нуклеотидных звеньев от 41 до 71, имеющие нуклеотидные последовательности SEQ ID NO: 1, 2, 3, 4, 5, обладающие способностью высокоаффинно связывать белок Dickkopf-1 человека, в том числе в образцах сыворотки крови пациентов с иммуновоспалительным ревматическим заболеванием. Изобретение обеспечивает повышение аффинности ДНК-аптамеров, специфично и прочно связывающихся с белком Dickkopf-1 человека, а также расширение ассортимента ДНК-аптамеров. 7 ил., 3 пр.
ДНК-аптамер, специфично связывающийся с белком Dickkopf-1 человека, полученный в результате селекции in vitro и представляющий собой олигодезоксирибонуклеотид, выбранный из группы:
ДНК-аптамер DK1 с длиной 71 нуклеотид, с молекулярной массой 21.9 кДа, с константой диссоциации 1.3±1 нМ, имеющий последовательность SEQ ID NO 1;
ДНК-аптамер DK2 с длиной 71 нуклеотид, с молекулярной массой 21.8 кДа, с константой диссоциации 1.8±1 нМ, имеющий последовательность SEQ ID NO 2;
ДНК-аптамер DK4 с длиной 71 нуклеотид, с молекулярной массой 21.9 кДа, с константой диссоциации 1.8±0.5 нМ, имеющий последовательность SEQ ID NO 3;
ДНК-аптамер DK4-41t с длиной 41 нуклеотид, с молекулярной массой 12.6 кДа, с константой диссоциации 5.6±3 нМ, имеющий последовательность SEQ ID NO 4;
ДНК-аптамер DK2-48t с длиной 48 нуклеотидов, с молекулярной массой 14.7 кДа, с константой диссоциации 3.3±2 нМ, имеющий последовательность SEQ ID NO 5.
| CN 109576272 A, 05.04.2019 | |||
| CN 107760686 А, 06.03.2018 | |||
| VOROBYEVA M | |||
| A | |||
| et al | |||
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
| Джино-прядильная машина | 1922 |
|
SU173A1 |
| Переносная печь для варки пищи и отопления в окопах, походных помещениях и т.п. | 1921 |
|
SU3A1 |
| Приспособление для обрезывания караваев теста | 1921 |
|
SU317A1 |
| СПОСОБ СЕЛЕКЦИИ АПТАМЕРОВ К ЗАДАННЫМ БЕЛКОВЫМ МИШЕНЯМ НА ПОВЕРХНОСТИ КЛЕТОК | 2012 |
|
RU2518368C1 |
Авторы
Даты
2024-03-01—Публикация
2023-07-28—Подача