Изобретение относится к области молекулярной генетики и может быть использовано для проведения амплификации четырех генов саркомерных белков (MYH7, TPM1, TNNT2 и TNNI3) с помощью метода ПЦР длинных фрагментов ДНК. Применение синтетических олигонуклеотидных проб позволяет выполнить таргетное «обогащение» полной нуклеотидной последовательности анализируемых генов саркомерных белков, что служит основой для получения информации об экзонных и интронных вариантах (однонуклеотидные, мелкие инсерции и делеции, крупные делеции) в генах саркомерных белков, патогенные варианты которых являются частой причиной развития гипертрофической кардиомиопатии [1, 2]. Особенность набора олигонуклеотидных проб заключается в получении длинных и перекрывающихся фрагментов ДНК при проведении ПЦР, что имеет важное значение для реконструкции индивидуальных гаплотипов у пациентов с гипертрофической кардиомиопатией, особенно в случае детекции нескольких патогенных вариантов в одном гене и отсутствии информации об их наследовании от родителей. Информация о вариабельности полной нуклеотидной последовательности генов саркомерных белков важна не только для диагностики гипертрофической кардиомиопатии, но и для характеристики частот аллелей и генотипов, фазирования генетических вариантов с определением их цис-/транс-положения и реконструкции структуры гаплотипов данных генов у индивидов в различных популяциях.
Внедрение методов высокопроизводительного секвенирования ДНК требует разработки методов «обогащения» образца тотальной ДНК человека таргетными (целевыми) регионами, так как для отдельных исследований чаще всего нужно определить последовательность конкретных генов. Наиболее близким решением является использование таргетных методов обогащения. Существенным недостатком известных методов оценки генетической компоненты гипертрофической кардиомиопатии является невозможность оценки глубоких интронных областей в генах, ответственных за развитие заболевания. Полногеномные методы диагностики, несмотря на возможность оценки интронных областей, имеют ряд недостатков и ограничений: во-первых, необходимо большое количество входной ДНК; во-вторых, это наиболее дорогостоящий подход к поиску генетических вариантов, ассоциированных с определенным заболеванием; в-третьих, объем получаемых данных является избыточным.
В настоящее время известны коммерческие наборы олигонуклеотидных проб, позволяющих провести амплификацию фрагментов ДНК и в последующем при секвенировании и биоинформатической обработке данных осуществить поиск вариантов в генах, ассоциированных с кардиологическими заболеваниями, в том числе с гипертрофической кардиомиопатией. Они предназначены для исследования экзонов и прилегающих к ним коротких участков интронов («TruSight™ Cardio Sequencing Panel», Illumina; «Comprehensive Cardiomyopathy NGS Panel», Fulgent Genetics). Существуют наборы олигонуклеотидных проб, которые используются для исследования всех (или большинства) белок-кодирующих генов человека (экзом), которые также направлены на исследование кодирующих участков для каждого гена: например, «Illumina DNA Prep with Exome 2.0 Plus Enrichments, Illumina; «SureSelect Human All Exon», Agilent. Кроме того, возможна разработка кастомных (пользовательских) генных панелей для высокопроизводительного секвенирования, основанных на мультиплексных ПЦР, в которых получаются ПНР-продукты, содержащие экзонные последовательности генов.
Интронная изменчивость генов в настоящее время может быть изучена, главным образом, в результате проведения полногеномного секвенирования, которое является дорогостоящим подходом. Вместе с тем, описаны случаи, когда причиной гипертрофической кардиомиопатии были интронные варианты, приводящие к нарушению нормального сплайсинга мРНК [4, 5]. В последнее время появились отдельные работы, в которых для таргетного обогащения используется ПЦР длинных фрагментов ДНК, однако они направлены на уменьшение числа ПЦР-продуктов в эксперименте через мультиплексирование и не ставят задачи получения полной последовательности гена и определения цис/транс-положения выявляемых генетических вариантов относительно друг друга [6, 7].
Технической задачей изобретения является подбор последовательности синтетических олигонуклеотидных проб для амплификации полной последовательности генов (MYH7,TPM1,TNNT2 и TNNI3), кодирующих белки саркомера сердечной мышцы.
Поставленная задача решена путем разработки синтетических олигонуклеотидных последовательностей, выступающих в качестве праймеров для целевых фрагментов анализируемых генов MYH7,TPM1,TNNT2 и TNNI3 и проведения ПЦР длинных фрагментов ДНК.
Техническим результатом изобретения является получение ПЦР-продуктов длиной от 4665 до 8749 пар оснований, перекрывающихся друг с другом в пределах одного гена при выравнивании на последовательность генома человека, предназначенных для дальнейшего секвенирования и исследования изменчивости генов саркомерных белков, наиболее часто вовлеченных в развитие гипертрофической кардиомиопатии.
Использование данного набора ведет к повышению точности диагностики причин развития гипертрофической кардиомиопатии по сравнению с существующими аналогами. Информация о вариабельности полной нуклеотидной последовательности генов саркомерных белков может быть использована для оценки особенностей частот аллелей и генотипов, фазирования генетических вариантов с определением их цис-/транс-положения и реконструкции структуры гаплотипов данных генов у индивидов в различных популяциях.
Сущность заявляемого технического решения поясняется графическими изображениями, на которых:
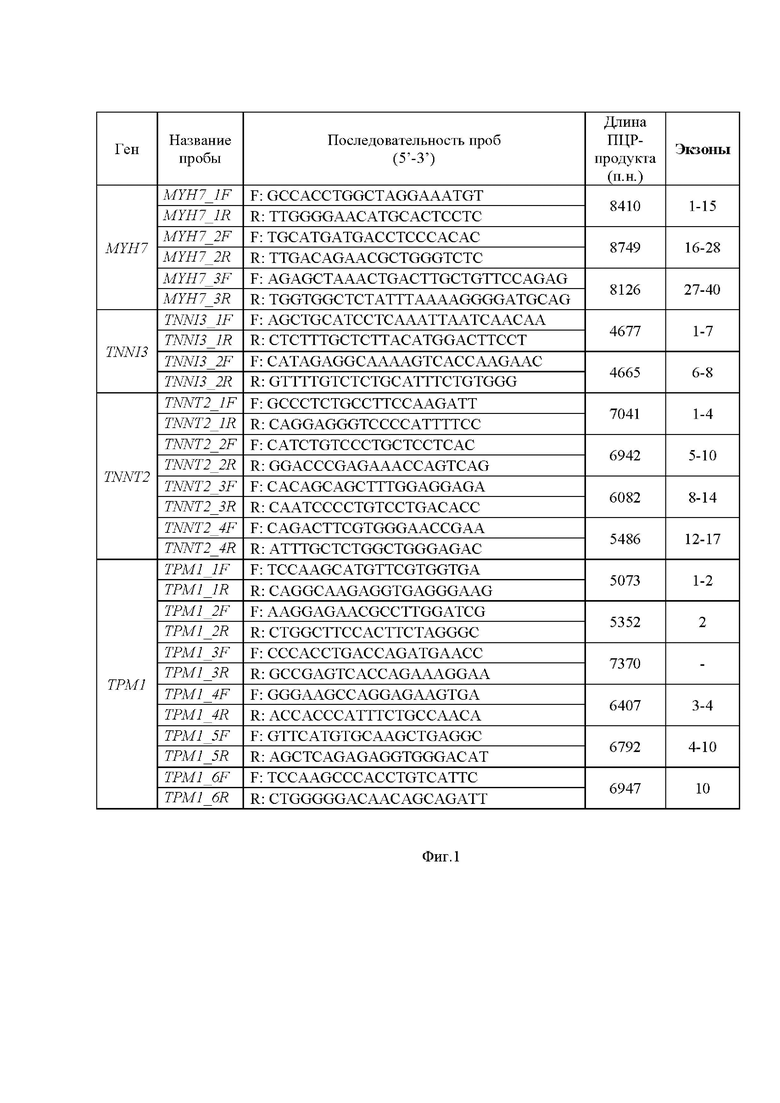
Фигура 1. Структура набора синтетических олигонуклеотидных проб.
Фигура 2. Схематическое изображение расположения олигонуклеотидных проб и размер ПЦР-продуктов в генах саркомерных белков MYH7, TPM1, TNNT2 и TNNI3.
Описание изобретения
В разработанном наборе синтетических олигонуклеотидных проб (праймеров) для четырех генов саркомерных белков MYH7, ТРМ1, TNNT2 и TNNI3, праймеры подобраны таким образом, чтобы все нарабатываемые фрагменты ДНК покрывали полную последовательность выбранных генов. Кроме того, получаемые ПЦР - продукты перекрываются между собой в пределах отдельных генов. Это свойство позволяет определять гаплотипы при условии наличия нескольких вариабельных сайтов (цис- или транс-положение вариантов относительно друг друга). Праймеры были протестированы на специфичность с помощью программы BLAST [3]. Концентрация рабочих растворов праймеров в реакционной смеси ПЦР составляет 10 пикомоль/мкл.
Амплификацию для каждого фрагмента ДНК с помощью соответствующих пар праймеров осуществляли по протоколу производителя наборов для амплификации ПЦР длинных фрагментов следующим образом:
1. В качестве матрицы ДНК для исследования используются образцы ДНК, полученные из биологических ядросодержащих тканей индивидов, выделенных любым общепринятым способом. Основным требованием к качеству образца ДНК является отсутствие каких-либо признаков его деградации, примесей РНК, высоких концентраций солей и белков, достаточная средняя длина фрагментов ДНК в образце (несколько т.п.о.). Качество образца ДНК оценивают с помощью электрофоретического разделения в 1,5% агарозном геле, а также с помощью спектрофотометра NanoDrop 2000 или его аналога при длинах волн 260 нм и 280 нм.
2. Образцы ДНК должны быть разведены до рабочей концентрации в пределах 10-100 нг/мкл.
3. Реакционная смесь для проведения ПЦР длинных фрагментов ДНК состоит из смеси буфера для полимеразы и полимеразы, согласно производителю набора, а также пары праймеров для фрагментов, покрывающих определенных участок генов саркомерных белков, образца ДНК и деионизованной воды. Общее количество ДНК для одной реакционной смеси должно составлять 200 нг. Реакционная смесь для проведения ПЦР длинных фрагментов должна содержать специальные ферменты и соотношение компонентов, необходимое для получения продуктов длиной несколько т.п.о. Использование реактивов различных производителей может потребовать оптимизации протокола ПЦР (длительности шагов и температурного режима).
4. Протокол программы амплификации основан на принципе стандартной трех шаговой ПЦР, включающей: этап денатурации при +94°С -+98°С, этап отжига праймеров при +60°С -+72°С и элонгации при +68°С -+72°С. Этап денатурации варьирует в пределах 20-30 сек., этап отжига длится 30 сек., этап элонгации длится из расчета (длина амплифицируемого фрагмента (т.п.о.)* 1 мин. +10 сек/цикл). Продолжительность этапов «предварительного нагрева» и «финальной элонгации» регламентируется протоколом производителя набора для ПЦР. Хранение готовых продуктов ПЦР проводится при от +4°С до +12°С, до суток. Выполнение протокола возможно на амплификаторах с подогреваемой крышкой и наличием функций градиента температур и времени.
Техническим результатом является получение ПЦР-продуктов длиной от 4665 до 8749 пар оснований, перекрывающихся друг с другом в пределах одного гена при выравнивании на последовательность генома человека (фиг. 1), предназначенных для дальнейшего секвенирования и исследования изменчивости генов саркомерных белков.
Полученные продукты ПЦР могут быть подвергнуты секвенированию на любой платформе для секвенирования ДНК, после приготовления соответствующих ДНК-библиотек, в том числе секвенированию «следующего поколения» (NGS), секвенированию «третьего поколения» (длинных единичных молекул), а также секвенированию по Сэнгеру (при условии подбора дополнительных праймеров для секвенирования).
Изобретение позволяет проводить поиск патогенных вариантов в генах саркомерных белков у пациентов с гипертрофической кардиомиопатией с помощью секвенирования ДНК, что в итоге приводит к более точной диагностике наследственных причин заболевания.
Данные о вариабельности полной нуклеотидной последовательности генов саркомерных белков могут быть использованы для оценки особенностей частот аллелей и генотипов, фазирования генетических вариантов с определением их цис-/транс-положения и реконструкции структуры гаплотипов данных генов у индивидов в различных популяциях.
Источники информации:
1. Ingles J, Goldstein J, Thaxton С, et al. Evaluating the Clinical Validity of Hypertrophic Cardiomyopathy Genes. Circ Genom Precis Med. 2019;12(2):e002460. doi: 10.1161/CIRCGEN. 119.002460
2. Gerull, В.; Klaassen, S.; Brodehl, A. The genetic landscape of cardiomyopathies. In Genetic Causes of Cardiac Disease; Erdmann, J., Moretti, A., Eds.; Springer: Cham, Switzerland, 2019; Vol. 7, pp. 45-91.
3. Ye J, Coulouris G, Zaretskaya I, Cutcutache I, Rozen S, Madden TL. Primer-BLAST: a tool to design target-specific primers for polymerase chain reaction. BMC Bioinformatics. 2012;13:134. doi: 10. 1186/1471-2105-13-134
4. Mendes de Almeida R, Tavares J, Martins S, et al. Whole gene sequencing identifies deep-intronic variants with potential functional impact in patients with hypertrophic cardiomyopathy. PLoS One. 2017;12(8):e0182946. doi: 10.1371/joumal. pone. 0182946
5. Xiong HY, Alipanahi B, Lee LJ, et al. RNA splicing. The human splicing code reveals new insights into the genetic determinants of disease. Science. 2015;347(6218):1254806. doi: 10.1126/science. 1254806
6. Leija-Salazar M, Sedlazeck FJ, Toffoli M, et al. Evaluation of the detection of GBA missense mutations and other variants using the Oxford Nanopore MinION. Mol Genet Genomic Med. 2019;7(3):e564. doi: 10.1002/mgg 3.564
7. Soufi M, Bedenbender S, Ruppert V, Kurt B, Schieffer B, Schaefer JR. Fast and Easy Nanopore Sequencing Workflow for Rapid Genetic Testing of Familial Hypercholesterolemia. Front Genet. 2022;13:836231. doi:10.3389/fgene.2022.836231
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ генотипирования полиморфного локуса rs139794067 (G>T) гена MYL3 | 2025 |
|
RU2837876C1 |
| НАБОР СПЕЦИФИЧЕСКИХ ГИБРИДИЗАЦИОННЫХ ЗОНДОВ ДЛЯ ОПРЕДЕЛЕНИЯ ИЗМЕНЕНИЙ В ГЕНЕ ЛИЗОСОМА-АССОЦИИРОВАННОГО ПРОТЕИНА-2 (LAMP2) У ЧЕЛОВЕКА МЕТОДОМ MLPA ДЛЯ ДИАГНОСТИКИ БОЛЕЗНИ ДАНОНА | 2021 |
|
RU2781084C1 |
| Набор синтетических олигонуклеотидов для определения последовательности 1 интрона, примыкающей ко 2 экзону, генов системы HLA I и II классов (HLA-A и HLA-DRB1) | 2016 |
|
RU2649064C1 |
| СПОСОБ ПОЛУЧЕНИЯ РЕКОМБИНАНТНОГО ИММУНОГЛОБУЛИНА IGA2m1-ИЗОТИПА В КЛЕТКАХ МЛЕКОПИТАЮЩИХ | 2022 |
|
RU2801178C1 |
| Способ конструирования минигенов млекопитающих и рекомбинантная плазмида pgC1HDR, кодирующая миниген ингибитора С1 эстеразы человека, предназначенная для получения гуманизированных по гену Serping1 мышей | 2022 |
|
RU2805177C1 |
| Способ анализа соматических мутаций в генах BRAF, NRAS и KIT с использованием LNA-блокирующей мультиплексной ПЦР и последующей гибридизацией с олигонуклеотидным биологическим микрочипом (биочипом) | 2017 |
|
RU2674338C1 |
| Способ получения димерной формы мутантного иммуноглобулина IgA2m1-изотипа в клетках млекопитающих | 2023 |
|
RU2822889C1 |
| Способ секвенирования экзонов гена HLA-DPA1 и набор синтетических олигонуклеотидов для его реализации | 2024 |
|
RU2837865C1 |
| Способ получения димерной формы иммуноглобулина IgA1-изотипа в клетках млекопитающих | 2023 |
|
RU2822890C1 |
| Способ пробоподготовки образцов изолятов коронавируса SARS-CoV-2 и олигонуклеотидные праймеры для его реализации | 2021 |
|
RU2762759C1 |

Изобретение относится к области молекулярной биологии. Описан набор синтетических олигонуклеотидных проб для проведения амплификации полной нуклеотидной последовательности генов саркомерных белков MYH7, TPM1, TNNT2 и TNNI3 методом ПЦР длинных фрагментов ДНК. Набор характеризуется тем, что при его использовании происходит таргетное обогащение полной нуклеотидной последовательности анализируемых генов, служащее основой для анализа вариантов на протяжении всей последовательности генов, в том числе интронов и нетранслируемых областей. Техническим результатом является получение ПЦР-продуктов длиной от 4665 до 8749 пар оснований, перекрывающихся друг с другом в пределах одного гена при выравнивании на последовательность генома человека, предназначенных для дальнейшего секвенирования и исследования изменчивости генов саркомерных белков. 2 ил.
Набор синтетических олигонуклеотидных проб для проведения амплификации полной нуклеотидной последовательности генов саркомерных белков MYH7, TPM1, TNNT2 и TNNI3 методом ПЦР длинных фрагментов ДНК, характеризующийся тем, что при его использовании происходит таргетное обогащение полной нуклеотидной последовательности анализируемых генов, служащее основой для анализа вариантов на протяжении всей последовательности генов, в том числе интронов и нетранслируемых областей, структура которых имеет следующие характеристики:
| НАБОР ПРАЙМЕРОВ ДЛЯ ДИАГНОСТИКИ НАСЛЕДСТВЕННОЙ КАРДИОМИОПАТИИ | 2019 |
|
RU2745079C1 |
| CN 109486937 A, 19.03.2019 | |||
| CN 102965428 A, 13.03.2013 | |||
| Кучер А.Н., Валиахметов Н.Р., Салахов Р.Р., Голубенко М.В., Павлюкова Е.Н., Назаренко М.С | |||
| Прибор для контроля непрерывности поездного тормозного трубопровода | 1925 |
|
SU870A1 |
| Способ получения продуктов конденсации фенолов с формальдегидом | 1924 |
|
SU2022A1 |
| Переносная печь для варки пищи и отопления в окопах, походных помещениях и т.п. | 1921 |
|
SU3A1 |
Авторы
Даты
2024-03-05—Публикация
2023-06-08—Подача