Изобретение относится к области медицины, к кардиологии и генетической диагностике, а именно к способам выявления специфических мутаций в генах с использованием аллель-специфической полимеразной цепной реакции (ПЦР). Предлагаемый метод может быть использован для выявления мутации NM_000258.3:c.170C>A, в гене, который связан с гипертрофической кардиомиопатией (ГКМП).
Гипертрофическая кардиомиопатия (ГКМП) - самая распространенная форма наследственных заболеваний сердца, которая характеризуется гипертрофией стенки, как правило, левого желудочка, и встречается в общей популяции с частотой 1:500.
Отсутствие эффективных методов терапии во многом объясняется тем, что патогенетические механизмы ГКМП недостаточно хорошо изучены. Известно, что до 60% случаев ГКМП являются наследственными и вызваны мутациями в генах, кодирующих преимущественно саркомерные белки. Около 80% мутаций были обнаружены в генах MYH7 и MYBPC3, кодирующих тяжелую цепь β-миозина и миозин-связывающий белок С. Примерно 12% случаев наследственной ГКМП обусловлены мутациями в генах TNNT2, TNNI3 и TPM1, кодирующих кардиальный тропонин Т, кардиальный тропонин I и α-тропомиозин [Дементьева Е.В. и др. Генетический анализ пациентов с гипертрофической кардиомиопатией, Клетки и гены, 2020].
Проведенное целевое секвенирование ДНК пациентов с ГКМП позволило выявить как спектр патогенетически значимых вариантов в основных генах ГКМП, так и ряд новых вариантов с предполагаемой патогенетической значимостью, характерных для пациентов с ГКМП из России. У пяти пациентов нами были выявлены варианты с доказанной патогенетической значимостью в генах MYBPC3 и MYH7, а также 37 потенциально патогенетически значимых вариантов в генах MYBPC3, MYH7, TNNT2, MYL2, MYL3, TPM1, ACTN2, LDB3, TTN, MYH6, TCAP, NEXN, TRIM63, VCL, включая несколько впервые выявленных вариантов в генах MYBPC3, MYH7, TPM1, NEXN, TTN. Общая частота выявленных в основных генах ГКМП патогенетически значимых вариантов в российской популяции составила 20%, что в два раза ниже, чем в среднем в мире. Проведенный клинико-генетический анализ позволил выявить небольшой вклад известных отдельных патогенетически значимых вариантов и генов в развитие ГКМП в российской популяции [Филатова Е.В. и др. Поиск новых генетических факторов, связанных с развитием гипертрофической кардиомиопатии в российской популяции, Медицинская генетика, 2020].
В результате исследования [Анализ мутации p.Ala57Asp гена MYL3, обуславливающей гипертрофическую кардиомиопатию в этносах Дагестана - наиболее частого, генетически детерминированного заболевания сердца / Г. М. Раджабова, О. А. Махачев, М. Н. Дибирова, М. О. Раджабов. - Текст : непосредственный // Материалы X всероссийской научно-практической конференции. Махачкала, 2023]. По данным исследований, замена p.Ala57Asp в гене MYL3, вызывающая развитие гипертрофической кардиомиопатии, зафиксирована среди населения Дагестана, тогда как среди этнических славян она не выявлена. Данный результат исследования, подчеркивает возможность связи данной мутации с уникальными особенностями различных этнических групп, что делает настоящее изобретение актуальным.
Существует метод полноэкзомного секвенирования, в панель которого входит ген MYL3 [PMID: 33288880]. Метод полноэкзомного секвенирования - это секвенирование всех белок-кодирующих генов человека (приблизительно 20000), а секвенирование экзома с клинической целью - это секвенирование порядка 5000 генов, про которые на настоящий момент известна ассоциация с генетическими болезнями или признаками. Экзом составляет всего ~1% от полного генома человека, но приблизительно 85% всех болезнетворных вариантов находятся именно в белок-кодирующих областях. С помощью технологии секвенирования экзома можно определить последовательность 90-95% целевых участков человека, и некоторые участки поддаются секвенированию с помощью этой методики несколько хуже. Способность метода выявить болезнетворный вариант зависит от того, в каком участке он находится (Online Mendelian Inheritance in Man, OMIM. http://www.ncbi.nlm.nih.gov/omim/). Метод предназначен для поиска однонуклеотидных замен в кодирующих участках генов человека. Некоторые другие типы генетических вариантов могут быть выявлены, но поддаются обнаружению с меньшей вероятностью, чем однонуклеотидные замены: это относится, в частности, к коротким делециям или вставкам (инделам) и изменениям числа коротких тандемных повторов (Database of Single Nucleotide Polymorphisms (dbSNP). http://www.ncbi.nlm.nih.gov/SNP/).
Поэтому данный метод имеет ряд существенных ограничений, таких как высокая стоимость проведения анализа, значительные временные затраты на получение и интерпретацию результатов, а также высокая частота ложноположительных результатов. Помимо этого, считается нецелесообразно использовать данный метод для обнаружения конкретной мутации.
Было продемонстрировано применение технологии мономолекулярного секвенирования для ДНК-диагностики гипертрофической кардиомиопатии [Салахов Р.Р. и др., Медицинская генетика, 2020]. Обследованы 12 пациентов с установленным диагнозом “гипертрофическая кардиомиопатия”: 9 женщин и 3 мужчин, в возрасте от 18 до 67 лет. ДНК-библиотека для секвенирования была приготовлена на основе продуктов Long-Range ПЦР, охватывающих полную последовательность генов MYH7, MYBPC3, TNNT2, TNNI3 и TPM1 (по протоколу “PCR barcoding amplicons (SQKLSK109)”). Секвенирование выполнено на платформе MinION компании Oxford Nanopore Technologies (UK). Выявленные генетические варианты были подтверждены с помощью секвенирования по Сэнгеру. Результаты. Получены данные о полной последовательности пяти основных генов саркомерных белков гипертрофической кардиомиопатии. Из восьми вариантов в генах MYH7, MYBPC3 и TNNT2, выявленных в результате мономолекулярного секвенирования и потенциально являющихся причиной заболевания, были подтверждены с помощью секвенирования по Сэнгеру лишь четыре (p.Arg243Cys, p.Tyr609Asn, p.Arg870His в гене MYH7 и p.Lys985Asn гене MYBPC3). Проведен каскадный скрининг патогенного варианта p.Arg870His в гене MYH7. Установлено наличие гетерозиготного носительства у одной из дочерей пробанда.
Таким образом, технология мономолекулярного секвенирования имеет потенциал для диагностики гипертрофической кардиомиопатии при условии повышения точности секвенирования ДНК, а также оптимизации и упрощения биоинформатических алгоритмов идентификации генетических вариантов.
Ближайшим аналогом изобретения является набор праймеров, предназначенных для секвенирования для диагностики наследственной кардиомиопатии (RU 2745079). Предлагаемый набор праймеров, включает в себя последовательности нуклеотидов гена, выбранного из группы, включающей LMNA, TNNT2, RYR2, TTN, DES, ТМЕМ43, SCN5A, MYL3, TNNC1, DSP, PLN, FLNC, PRKAG2, MYPN, VCL, RBM20, BAG3, CSRP3, MYBPC3, PKP2, MYL2, PTPN11, MYH7, TGFB3, АСТС1, TPM1, JUP, DSC2, TTR, TNNI3, DMD, GLA, LAMP2, TAZ. Однако, для поиска конкретного полиморфизма нецелесообразно запускать диагностику всех наследственных кардиомиопатий.
В настоящее время, аллель-специфическая ПЦР - это эффективный метод выявления мутаций в геномной ДНК, широко используемый для анализа однонуклеотидных полиморфизмов (SNPs). Так как этот метод позволяет найти небольшое количество мутантных ДНК среди большого количества ДНК дикого типа, он также применяется для анализа соматических мутаций.
В основе метода лежит элонгация аллель-специфических праймеров (allele specific primer extension), строго комплементарных нуклеотидной последовательности матричной ДНК, расположенной на 3'-конце. При неполной комплементарности эффективность удлинения праймера во время ПЦР резко снижается либо отсутствует.
Преимущества метода заключаются в его специфичности, чувствительности, невысокой стоимости и быстроте получения результатов, так как при правильно подобранных условиях анализ SNPs занимает всего 3-4 часа. Он позволяет с высокой точностью различать аллели в геноме в одностадийной ПЦР без дополнительного этапа разделения продуктов реакции.
Выдвигаемая техническая проблема, на разрешение которой направлено заявляемое изобретение, является разработка эффективного и быстрого способа обнаружения варианта NM_000258.3:c.170C>A (p.Ala57Asp) в экзоне 3 гена MYL3.
Разработан способ генотипирования полиморфного локуса rs139794067 (G>T) гена MYL3 методом аллель-специфической ПЦР с последующим электрофорезом продуктов амплификации, отличающийся тем, что идентификацию аллельных вариантов осуществляют с использованием прямого и обратного праймера для аллеля дикого типа - SEQ ID NO 1 и SEQ ID NO 3, для мутантного SEQ ID NO 2 и SEQ ID NO 3, соответственно.
Техническим результатом заявленного изобретения является создание новой аллель-специфической ПЦР, позволяющей точно и быстро идентифицировать мутацию NM_000258.3:c.170C>A (p.Ala57Asp) в экзоне 3 гена MYL3. Заявленный способ позволяет точно идентифицировать наличие как аллелей дикого типа, так и мутантных аллелей гена MYL3 на основе специфичности разработанных уникальных праймеров SEQ ID NO 1 - SEQ ID NO 3, что дает возможность широко использовать его в молекулярно-генетической диагностике.
В разработанном способе использует обычную амплификацию фрагментов ДНК по протоколу стандартной ПЦР с последующим анализом продуктов стандартным методом электрофореза в агарозном геле, что сильно удешевляет диагностику.
Использование данного метода значительно снижает так же и денежные затраты, что делает диагностику более экономически выгодной по сравнению с другими известными методами. Для проведения аллель-специфической ПЦР не требуется сложного оборудования или специализированных навыков, что делает метод доступным для большинства лабораторий. Метод требует минимального количества биологического материала, что особенно важно при работе с ограниченными образцами.
КРАТКОЕ ОПИСАНИЕ ГРАФИЧЕСКИХ МАТЕРИАЛОВ
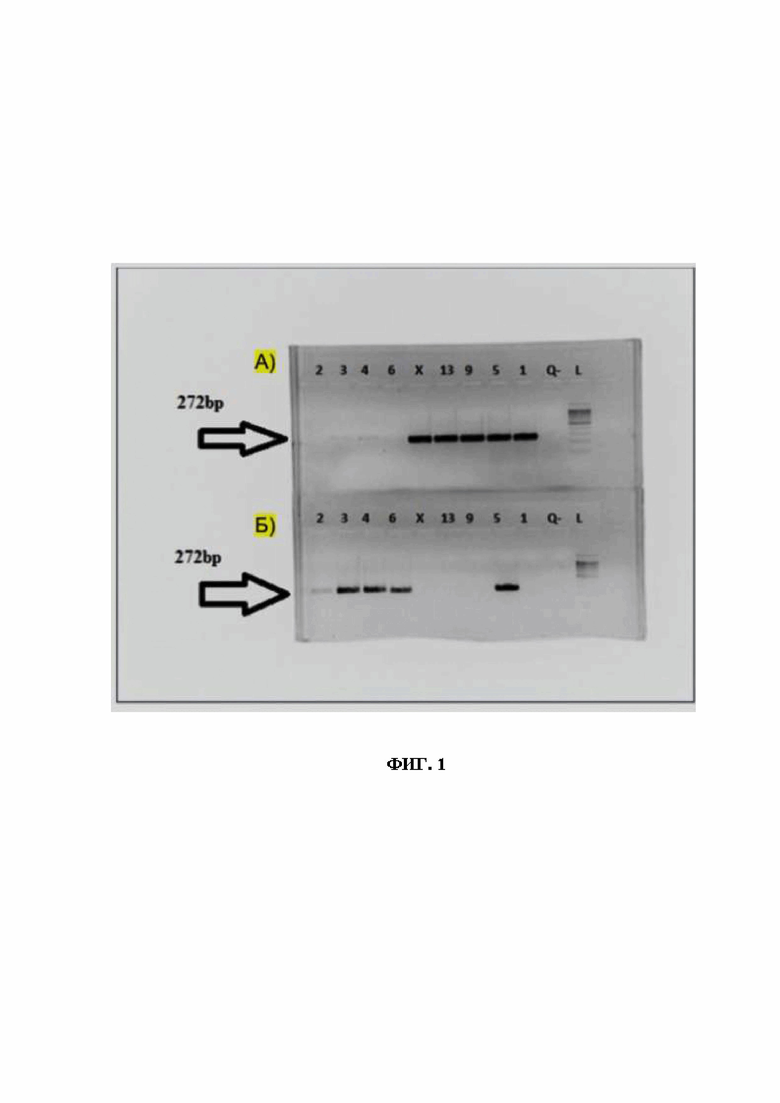
ФИГ. 1. Электрофореграммы результатов аллель-специфической ПЦР:
А) размер фрагмента на праймерах MYL3-WT+Rev на норму длиной 272 bp; Б) размер фрагмента на праймерах MYL3-Mut+Rev длиной 272 bp. Q- - отрицательный контроль ПЦР (отсутствие матрицы ДНК). L - маркер молекулярной массы ДНК (100-1000bp).
ОСУЩЕСТВЛЕНИЕ ИЗОБРЕТЕНИЯ
Сущность технического решения заключается в проведении амплификации в двух параллельных пробирках, содержащих различные пары праймеров. В первой пробирке используется пара праймеров, специфичных к участку гена MYL3, содержащему неизмененный (дикий тип, WT) аллель. Во второй пробирке применяется пара праймеров, направленных на амплификацию участка гена, содержащего мутацию. Такой подход позволяет одновременно детектировать наличие мутации, а также устанавливать генотип пациента.
Способ осуществляют следующим образом:
1. Извлечение ДНК из биологического образца (крови, слюны, тканей и т.д.) с контролем концентрации на спектрофотометре.
2. Приготовление реакционных смесей для ПЦР, включающих специфические праймеры, комплементарные участку гена MYL3, который потенциально содержит мутацию NM_000258.3:c.170C>A (p.Ala57Asp).
3. Проведение ас-ПЦР с использованием праймеров, позволяющих амплифицировать только мутантный аллель в одной смеси, только нормальный аллель во второй смеси.
4. Анализ результатов ПЦР с использованием электрофореза или других методов детекции.
Согласно настоящему изобретению, технический эффект достигается за счет разработки уникальных условий, протоколов и специфических праймеров (Таблица 1), предназначенных для проведения аллель-специфической полимеразной цепной реакции с целью выявления мутации NM_000258.3:c.170C>A (p.Ala57Asp) в экзоне 3 гена MYL3.
Реакционная смесь для проведения аллель-специфической ПЦР включает следующие компоненты:
1. ДНК-матрица - 1мкл;
2. Праймер MYL3-Rev - 1мкл;
3. Праймер MYL3-WT (F) (для второй смеси MYL3-Mut (F)) - 1мкл;
4. 10xSNP-буфер (MgCl2 free) - 2мкл;
5. H2O, nuclease-free - 8,5 мкл;
6. Смесь dNTP (10mM) - 0,5мкл.
7. MgCl2 (50mM) - 1мкл;
8. SNP-полимераза (5 ед.\мкл) - 0,2мкл
Экспериментально подобраны оптимальные условия для проведения генотипирования, амплификация проводится в термоциклере с использованием следующих этапов:
1. Денатурация - 95°C - 3 мин;
2. Циклирование - 38 циклов - 94°C, 30 сек - 62°C, 30 сек - 72°C ,1 мин;
3. Финальная элонгация - 72°C - 2 мин;
4. Охлаждение - до 4-10°C.
С применением открытого ресурса University of California Santa Cruz (UCSC) [https://genome.ucsc.edu/index.html] выбран сиквенс, фланкирующий искомую однонуклеотидную замену [G>T] rs139794067 гена MYL3, и затем с помощью доступного онлайн програмного обеспечения OligoAnalize [https://eu.idtdna.com/pages/tools/oligoanalyzer] подобрана последовательность олигонуклеотидов, используемых для проведения ПЦР-реакции (таблица 1).
Было обследовано 126 пациентов.
Таблица 1.
Набор олигонуклеотидных праймеров.
Для идентификации аллелей используются два типа праймеров:
Обратный праймер (MYL3-Rev): CGGGACAATGCGAGATG, который остается неизменным для обоих аллелей (SEQ ID NO 3).
Прямые праймеры:
- для выявления аллеля дикого типа (G): MYL3-WT (F) GGCAGAGTTCAAGGAAGC (SEQ ID NO 1);
- для амплификации мутантного аллеля (T): MYL3-Mut (F) GGCAGAGTTCAAGGAAGA (SEQ ID NO 2);
Длина целевого фрагмента 272 п.н. в обоих случаях.
После амплификации проводят электрофорез в 2%-ном агарозном геле с последующим анализом полученных данных.
Пример полученного результата показан на фигуре 1: образцы 2, 3, 4, 6 являются гомозиготными носителями варианта, образец 5 является гетерозиготным носителем варианта. X, 13, 9, 1 - замены нет).
Таким образом, если мы видим на агарозе результат только амплификации первой пары, тогда пациент гомозигота норма, если обеих пар, то гетерозигота, если только второй пары, то гомозигота A57V.
ПРИМЕР 1.
Пациент М. 38 лет, пол - мужской. Диагноз: Гипертрофическая кардиомиопатия. Исследование: панель генов «Наследственные заболевания сердца и сосудов».
Методом секвенирования нового поколения было проведено секвенирование белок-кодирующих генов образца ДНК и проведен биоинформатический поиск в панели ассоциированных генов. Выявлен вероятно патогенный генетический вариант NM_000258.3:c.170C>A (p.Ala57Asp) в гомозиготном состоянии в последовательности 3 экзона гена MYL3.
Обнаруженный генетический вариант был проверен с использованием настоящего изобретения на основе аллель-специфичных полимеразных цепных реакций.
Выделение ДНК из крови проводили колоночным методом следующим образом. На первом этапе был отобран лейкоцитарный слой. Далее клетки были пролизированы с помощью специального буфера и протаиназы К (набор для выделения Magen). Далее добавляли спирт и переносили весь объем на колонку. После термостатирования при комнатной температуре (5 мин) производили центрифугирование в течение 1 минуты при 20000 rcf. После этого колонку чистили двухэтапно промывочными растворами с чередующим центрифугированием в течении 1 минуты при 10000 rcf. С полученной чистой колонки элюировали ДНК 60 мкл элюирующим буфером. Последний этап провели дважды для увелечения выхода продукта.
Подготовка образцов ДНК к генотипированию. Качество выделенной ДНК оценивали по степени чистоты и концентрации раствора на спектрофотометре NanoDrop (Thermo Fisher Scientific, США). Все анализируемые образцы ДНК были разведены деионизированной водой до концентрации 15-20 нг/мкл при А260/280=1,5-2,0.
Аллель-специфическую ПЦР проводили в 25 мкл двух реакционных смесей, содержащей 4. 10xSNP-буфер (MgCl2 free) («Евроген», Москва, Россия), смесь dNTP 10mM («Евроген», Москва, Россия), MgCl2 50mM («Евроген», Москва, Россия), SNP-полимераза 5 ед.\мкл ((«Евроген», Москва, Россия), 15 нг ДНК, по 1 мкМ каждого праймера (SEQ ID NO 1 и SEQ ID NO 3 - для первой смеси, SEQ ID NO 2 и SEQ ID NO 3 - для второй смеси), 8,5 мкл H2O, nuclease-free. Реакция амплификации состояла из стадии нагревания до 95°C в течение 3 минут, затем 38 циклов [94°C в течение 30 секунд и 62°C в течение 30 секунд, 72°C в течение 1 минуты], затем финальная элонгация при 72°C в течение 2 минут, охлаждение для хранения до 4°C.
После проведения аллель-специфической реакции генотипирование осуществляли по данным гель-электрофореза. Для аллеля дикого типа результат анализируется, где были задействованы праймеры SEQ ID NO 1 и SEQ ID NO 3, для мутантного, соответственно, SEQ ID NO 2 и SEQ ID NO 3.
ПРИМЕР 2.
Пациент Т. 25 лет, пол - мужской. Диагноз: Внезапная сердечная смерть в семье, мыш.гипотрофия? Исследование: поиск генетического варианта NM_000258.3:c.170C>A (p.Ala57Asp) в экзоне 3 гена MYL3.
Методика выполнения исследования как в примере 1.
Генетический вариант был проверен у пациента Т. с использованием настоящего изобретения на основе аллель-специфичных полимеразных цепных реакций. Результаты исследования: генетический вариант NM_000258.3:c.170C>A (p.Ala57Asp) в гомозиготном состоянии в последовательности 3 экзона гена MYL3 выявлен у пациента Т.
Таким образом, предложенный способ идентификации генотипирования полиморфного локуса rs139794067 (G>T) гена осуществляют посредством проведения аллель-специфической ПЦР в присутствии разработанных уникальных специализированных олигонуклеотидных праймеров, соответствующих мутантным и нормальным аллелям гена MYL3.
В разработанном способе использует обычную амплификацию фрагментов ДНК по протоколу стандартной ПЦР с последующим анализом продуктов стандартным методом электрофореза в агарозном геле, что сильно удешевляет диагностику.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="MYL3.xml"
softwareName="WIPO Sequence" softwareVersion="2.3.0"
productionDate="2025-02-25">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText></ApplicationNumberText>
<FilingDate>2025-02-25</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>20250225</ApplicantFileReference>
<ApplicantName languageCode="ru">ФГБНУ РНЦХ им. акад. Б.В.
Петровского</ApplicantName>
<ApplicantNameLatin>Petrovsky National Research Centre of
Surgery</ApplicantNameLatin>
<InventorName languageCode="ru">Заклязьминская Елена
Валерьевна</InventorName>
<InventorNameLatin>Zaklyazminskaya E.V.</InventorNameLatin>
<InventionTitle languageCode="ru">Способ генотипирования
полиморфного локуса rs139794067 (G>T) гена MYL3</InventionTitle>
<SequenceTotalQuantity>3</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggcagagttcaaggaagc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggcagagttcaaggaaga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>17</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgggacaatgcgagatg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
Изобретение относится к области молекулярной биологии. Описан способ генотипирования полиморфного локуса rs139794067 (G>T) гена MYL3 методом аллель-специфической ПЦР с последующим электрофорезом продуктов амплификации. Идентификацию аллельных вариантов осуществляют с использованием прямого и обратного праймеров: для аллеля дикого типа - SEQ ID NO: 1 и SEQ ID NO: 3, для мутантного SEQ ID NO: 2 и SEQ ID NO: 3 соответственно. Техническим результатом заявленного изобретения является создание новой аллель-специфической ПЦР, позволяющей точно и быстро идентифицировать мутацию NM_000258.3:c.170C>A (p.Ala57Asp) в экзоне 3 гена MYL3. 1 ил., 1 табл., 2 пр.
Способ генотипирования полиморфного локуса rs139794067 (G>T) гена MYL3 методом аллель-специфической ПЦР с последующим электрофорезом продуктов амплификации, отличающийся тем, что идентификацию аллельных вариантов осуществляют с использованием прямого и обратного праймеров: для аллеля дикого типа - SEQ ID NO: 1 и SEQ ID NO: 3, для мутантного SEQ ID NO: 2 и SEQ ID NO: 3 соответственно.
| CN 111411149 A, 14.07.2020 | |||
| НАБОР ПРАЙМЕРОВ ДЛЯ ДИАГНОСТИКИ НАСЛЕДСТВЕННОЙ КАРДИОМИОПАТИИ | 2019 |
|
RU2745079C1 |
| Osborn D | |||
| P | |||
| S | |||
| et al | |||
| Переносная печь для варки пищи и отопления в окопах, походных помещениях и т.п. | 1921 |
|
SU3A1 |
Авторы
Даты
2025-04-07—Публикация
2025-02-25—Подача