Предлагаемое изобретение относится к области медицинской микробиологии, а именно к способам молекулярно-генетического типирования штаммов Pseudomonas aeruginosa, которое может быть использовано при эпидемиологической диагностике внутрибольничных гнойно-септических, нозокомиальных инфекций на основе внутривидового типирования возбудителя.
В настоящее время P. aeruginosa является третьим наиболее часто выделяемым патогеном среди грамотрицательных палочек и седьмым среди всех микробных агентов, способных вызывать нозокомиальные инфекции. При этом на фоне эпидемии Covid - 19 данный этиологический агент продемонстрировал высокие показатели летальности среди пациентов с бактеримией (1, 2). Знание о клональной природе штаммов позволяет не только проводить противоэпидемические мероприятия, но и выявлять штаммы ранее не известные, поэтому необходимо использовать современные методы молекулярного типирования.
Несмотря на то, что существуют современные методы идентификации P. aeruginosa - ПЦР, серотипирование, сиквенирование в Российской Федерации на сегодняшний день, в нормативно-методической документации преобладают бактериологические методы идентификации синегнойной палочки (3,4,5).
Известное INDEL - типирование основано на определении «вставок-делеций» (INsertion-DELetion) в различных генах, что позволяет выявлять индивидуальные различия между штаммами микроорганизмов (6). По этому методу у искомого гена есть в наличии всего лишь два аллеля («вставка» и «делеция») указанный способ является гораздо проще остальных методов типирования, который был опробован на микроорганизмах V. cholerae (7), В. melitensis(8), Н. pylori (9). В связи с этим возник вопрос о создании нового способа внутривидового типирования и дифференциации для синегнойной палочки.
Известен способ типирования P. aeruginosa, основанный на применении серологических сывороток, используя которые возможно по структуре О-антигена разделять штаммы синегнойной палочки на 20 серогрупп (10,11). Их недостатком является, сложность получения, наличие чистой культуры для точности проведения анализа, ограниченность распространения сывороток для диагностики.
. Известны молекулярные методы типирования P. aeruginosa, как гель-электрофорез в пульсирующем поле, пульс-электрофореза в контролируемом гомогенном электрическом поле, MLST - типирование, VNTR-анализ (12,13,14,15,16). Однако недостатками этих методов является время выполнения анализа, достоверность, зависимость от иностранных баз данных, высокие затраты для проведения анализа, использование иностранного оборудования.
Наиболее близким методом к предлагаемому изобретению является способ генетического типирования штаммов P. aeruginosa за счет выявления SNP/INDEL, основанный на полногеномном секвенировании (17).
Однако данный способ имеет ряд недостатков. Анализ данных проводился с использованием полногеномных последовательностей, в которых сравнивались лишь парные изоляты от нескольких пациентов, между которыми сравнивались Indel-ассоциированные полиморфизмы, по которым определялись факторы фенотипической вариации. Получение полногеномных сиквенсов требует наличия дорогостоящего ДНК-секвенатора, а также использования дорогостоящих импортных расходных материалов, работы квалифицированного персонала.
Кроме того продолжительным и сложным остается анализ и интерпретация результатов, от десятков часов до нескольких дней с использованием высокопроизводительной техники и дорогостоящего программного обеспечения. Что в совокупности приводит к ограничению применения и высокой стоимости анализа.
Технической задачей предполагаемого изобретения является разработка нового способа позволяющего достоверно, быстро и с низкой себестоимостью осуществлять внутривидовую дифференциацию штаммов Р. aeruginosa.
Поставленная задача достигается тем, что в известном способе дифференциации штаммов Pseudomonas aeruginosa с помощью INDEL типирования, включающем выделение ДНК из исследуемого штамма Р. aeruginosa, постановку ПЦР со специфическими праймерами и учет реакции с помощью электрофореза, отличие состоит в том, что из ДНК исследуемого штамма выявляют четыре общих INDEL-генов, имеющие делеции определенного размера, а именно IND-РА0168, РА3324, РА0659 и РА1451, с последующей их амплификацией с помощью сконструированных специфических праймеров, выявляющих два альтернативных аллеля:
к гену IND-PA0168 - праймеры
AGGACATGGACTGCGTGG и
CCTGCTGCAGGCACTTGG
с длиной амплифицированного фрагмента 100 п. н. или 114 п. н.;
к гену IND-РА3324 - праймеры
GAGTCCCGGATGTGGTAGTC и
GATCACGCTCCACAGGTTGA
с длиной амплифицированного фрагмента 99 п. н. или 113 п. н.;
к гену IND-РА0659 - праймеры
GCGGCATCATCATCGAGAAG и
CTGGGCGGTGTGTAGGGA
с длиной амплифицированного фрагмента 106 п. н. или 112 п. н.;
к гену IND-РА1451 - праймеры
CAATTTCAGCGCCGGCGG и
CGAAGACGAACTGGACGAAC
с длиной амплифицированного фрагмента 100 п. н. или 109 п. н., при этом учет результатов дифференциации проводят визуально после электрофореза в 8% полиакриламидном геле в присутствии маркера молекулярных масс ДНК, причем для каждого штамма устанавливают уникальный INDEL-генотип по четырем INDEL-генам, а путем сравнения выявленных INDEL-генотипов между собой и с известными генотипами идентификационной таблицы устанавливают общее или различное происхождение исследуемых штаммов P.aeruginosa, позволяя дифференцировать один штамм от другого.
При этом ПЦР проводят в объеме 10 мкл и реакционная смесь содержит:
1,5 мМ MgCl, 0,2 мМ смеси дНТФ,
1,0 мкМ смеси праймеров (по 0,5 мкМ каждого праймера), 25 нг ДНК матрицы, 1 ед. ДНК-полимеразы, оставшийся объем - вода, при этом в качестве матрицы используют геномную ДНК объемом 5 мкл, которую получают из разных штаммов P. aeruginosa.
Кроме того ПЦР проводят с соблюдением режимов:
1 этап - денатурация при 94°С -10 с;
2 этап - отжиг при 60°С - 10 с;
3 этап - синтез при 72°С - 15 с (39 циклов)
Обоснование выбора праймеров.
С помощью программного обеспечения, разработанного авторами (ФКУЗ Ростовский-на-Дону противочумный институт Роспотребнадзора) было проанализировано более 5000 генов P. aeruginosa в базе данных GenBank. В процессе анализа нуклеотидных последовательностей штаммов P. aeruginosa в базе данных GenBank был определен ряд INDEL-генов, отличающихся по размеру у различных штаммов P. aeruginosa, и несущих только два аллельных варианта, с различными длинами ампликонов. По итогу анализа было выделено 4 общих INDEL-гена, формирующих продукты амплификации удобные для учета.
С помощью программного обеспечения BLAST и Primer - BLAST к вариабельным участкам выбранных генов РА0168, РА3324, РА0659, РА1451 были сконструированы специфические INDEL праймеры, смотри идентификационную таблицу 1.

Значения размеров амплифицированных фрагментов для каждого штамма по каждому из четырех INDEL-генов являются его индивидуальной характеристикой, что в свою очередь и позволяет дифференцировать один штамм от другого и определять их происхождение при помощи кластерного анализа.
Способ осуществляется следующим образом.
Перед осуществлением типирования штаммов P. aeruginosa выделяют ДНК исследуемой культуры. Бактериальную суспензию исследуемого штамма вносят в микропробирку объемом 1,5 мл, после чего проводят обеззараживание материала и выделение нуклеиновых кислот (ДНК) согласно МУ 1.3.2569-09 (18). Затем в четырех пробирках проводят амплификацию выделенной ДНК со специфическими праймерами.
Условия проведения реакции амплификации
Амплификацию проводят по следующей схеме: денатурация при 94°С -10 с, отжиг при 63°С - 10 с, синтез при 72°С - 15 с (39 циклов).
Реакционную смесь объемом 10 мкл готовят из расчета: 1,5 мМ MgCl, 0,2 мМ смеси дНТФ, 1,0 мкМ смеси праймеров (по 0,5 мкМ каждого праймера) 25 нг ДНКматрицы, 1 ед. ДНК-полимеразы, оставшийся объем -вода. В качестве матрицы используют геномную ДНК (объемом 5 мкл), полученную из разных штаммов P. aeruginosa. Учет результатов амплификации проводят с помощью электрофореза в 8% полиакриламидном геле в присутствии маркера молекулярных масс ДНК.
Учет результатов типирования проводят визуально после электрофореза, причем для каждого штамма устанавливают уникальный INDEL-генотип по четырем INDEL-генам, а путем сравнения выявленных NDEL-генотипов между собой и с известными генотипами идентификационной таблицы, устанавливают общее или различное происхождение исследуемых штаммов P. aeruginosa, что дает возможность дифференцировать один штамм от другого.
Общее время проведения исследования не превышает 9-12 часов.
Экспериментальные исследования подтверждают, что использование разработанного способа молекулярно-генетического типирования штаммов P. aeruginosa по структуре INDEL-генов, позволяет выявлять штаммы с их различными аллельными вариантами.
Таким образом, предложенный способ позволяет достоверно и быстро в течение 9-12 часов, проводить анализ штаммов P. aeruginosa, выделенных в различных странах, областях, регионах.
Пример 1.
В эксперименте использованы штаммы P. aeruginosa из коллекции Ростовского-на-Дону научно-исследовательского противочумного института Роспотребнадзора, выделенные в ДНР (2350, 2424, 2630, 2632) ив г. Хабаровске (21-10, 21-122, 21-454, 21-52). P. aeruginosa суспендировали в 100 мкл деионизованной воды, не содержащей нуклеаз. Далее проводят выделение ДНК согласно МУ 1.3.2569-09 (10). Для постановки полимеразной цепной реакции используют специфичные праймеры к INDEL-генам РА0168, РА3324, РА0659 и РА 1451.
Реакционную ПЦР смесь объемом 10 мкл готовят из расчета: 1,5 мМ MgCl, 0,2 мМ смеси дНТФ, 1,0 мкМ смеси праймеров (по 0,5 мкМ каждого праймера) 25 нг ДНКматрицы, 1 ед. ДНК-полимеразы, 5 микролитров ДНК, оставшийся объем - вода, после закапывали 1 каплю вазелинового масла. Смесь перемешивают и амплифицируют при условиях: денатурация при 94°С - 10 с, отжиг при 60°С - 10 с, синтез при 72°С - 15 с (39 циклов) в амплификаторе Терцик. Учет результатов амплификации проводят с помощью электрофореза в 8% полиакриламидном геле.
В таблице 2 показаны результаты электрофореза продуктов амплификации INDEL-генов: РА0168, РА3324, РА0659, РА1451.
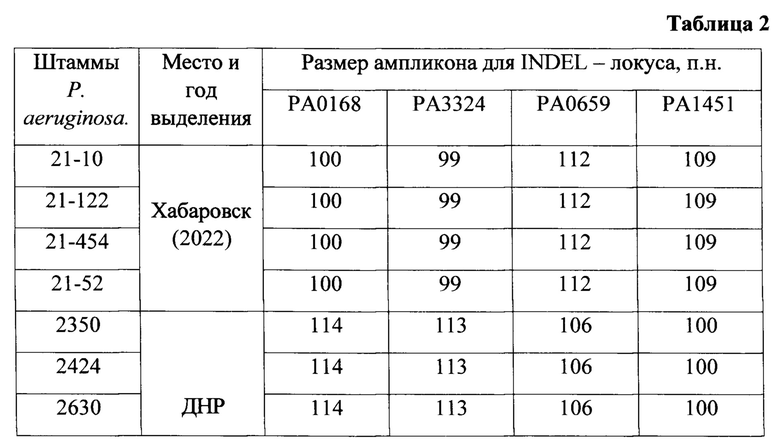

Вывод: Анализ результата амплификации INDEL-генов РА0168, РА3324, РА0659, РА1451, продемонстрировал, что значительная часть штаммов, выделенных в ДНР, отличаются по 4 INDEL - локусам от штаммов, выделенных в Хабаровске. Таким образом, изучаемые штаммы можно разделить на две большие группы. Следовательно, на этих территориях распространены генетически разные клоны. При этом в ДНР циркулируют преимущественно штаммы с набором аллелей: РА0168 - 114 п. н, РА3324 - 113 п. н, РА0659 - 106 п. н, РА1451 - 100 п. н. а в Хабаровске с набором аллелей: РА0168 - 100 п. н, РА3324 - 99 п. н, РА0659 - 112 п. н, РА 1451 - 109 п. н.
Пример 2.
В эксперименте использованы штаммы P. aeruginosa из коллекции Ростовского-на - Дону научно-исследовательского противочумного института, выделенные в ДНР (2679, 2889, 2911) и г. Ростов-на-Дону (39926, 45048, 45528). Условия проведения см
Пример 1.
В таблице 3 предоставлены результаты электрофореза продуктов амплификации INDEL-генов: РА0168, РА3324, РА0659, РА1451.
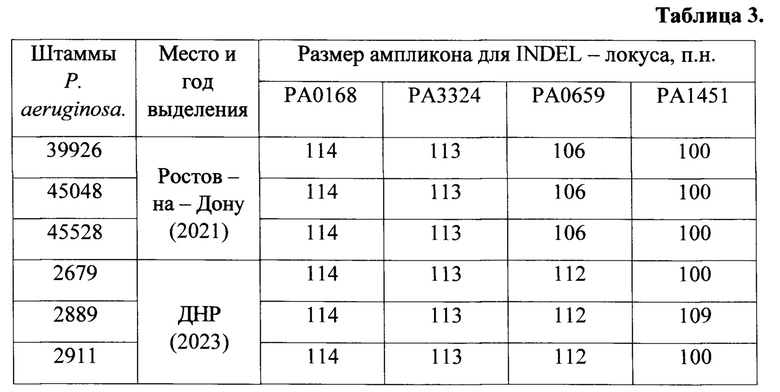
Таким образом анализ продуктов амплификации INDEL-генов РА0168, РА3324, РА0659, РА1451, выявил генетическое сходство штаммов по трем INDEL-локусам (РА0168, РА3324, РА1451), выделенных как на территории Ростова-на-Дону, так и на территории ДЫР. Однако у штамма 2889 есть отличие по длине аллельного варианта гена РА 1451 - 109 п. н. Различие штаммов выделенных в двух разных регионах идентифицируют по размеру алельного варианта гена РА0659, у штаммов из Ростова-на-Дону он составляет 106 п. н, а у штаммов из ДНР 112 п. н..
Вывод: на данных территориях циркулируют штаммы P. aeruginosa, которые можно разделить на две группы по INDEL-гену РА0659, отличающиеся от Хабаровска. Штамм 2889 будет входить в общую группу штаммов ДНР, и являтется обособленным генотипом.
Использование предполагаемого изобретения позволяет достоверно и быстро за счет сконструированных праймеров унифицировать и создать набор значений размера фрагментов аллелей для каждого штамма Р. aeruginosa по каждому из четырех INDEL-генов. Найденные INDEL-гены позволяют дифференцировать штаммы и определять их генетическое родство.
Предложенный способ дает возможность экономически выгодно проводить анализ штаммов P. aeruginosa, выделенных в различных странах, областях, регионах. К тому же это хорошее дополнение к молекулярной дифференциации штаммов с помощью полногеномного секвенирования.
Источники информации.
1. Малыгин А.С. и др. «ИССЛЕДОВАНИЕ СЛУЧАЕВ БАКТЕРИЕМИИ PSEUDOMONAS AERUGINOSA У БОЛЬНЫХ С ТЯЖЕЛЫМ ТЕЧЕНИЕМ COVID-19 », /Инфекционные болезни: Новости. Мнения. Обучение. - 2022. - Т. 11. - №. 4 (43). - С.47-55.
2. Sadeeva Z. Z. et al. Characterization of Pseudomonas aeruginosa isolated from positive samples of hemocultures and cerebrospinal fluid of children //Journal of microbiology, epidemiology and immunobiology. - 2022. - T. 99. - №. 3. - C. 309-321.
3. MP Методические рекомендации по бактериологической диагностике. синегнойной инфекции. М., 1984.
4. 6. МУК 4.2.2942-11. Методы санитарно-бактериологических исследований объектов окружающей среды, воздуха и контроля стерильности в лечебных организациях.
5. ГОСТ Р 54755-2011. Продукты пищевые. Методы выявления и определения количества бактерий вида Pseudomonas aeruginosa
6. Водопьянов, А. С., Водопьянов, С. О., Олейников, И. П., Мишанькин, Б. Н. INDEL-типирование штаммов Vibriocholerae //Эпидемиология и инфекционные болезни. - 2017. - Т. 22. - №. 4. - С.195-200.
7. «Способ молекулярно-генетического внутривидового типирования V. cholerae 01 и 0139 серогрупп », патент №2575046, кл. C12N 15/10, опубл. 10.02.2016 г. Бюл.№4.
8. «Способ генетического INDEL-типирования штаммов Brucella melitensis», патент №2732425,кл. C12Q 1/68, опубл. 16.09.2020 г. Бюл.№26
9. «Способ дифференциации штаммов Helicobacter pylori путем молекулярно-генетического типирования», патент №2688434, C12N 15/10, опубл.21.05.2019 Бюл. 15.
10. Brokopp С. D., Gomez-Lus R., Farmer III J. J. Serological typing of Pseudomonas aeruginosa: Use of commerical antisera and live antigens //Journal of Clinical Microbiology. - 1977. - T. 5. - №. 6. - C. 640-649.
11. Huszczynski S. M., Lam J. S., Khursigara С.M. The role of Pseudomonas aeruginosa lipopolysaccharide in bacterial pathogenesis and physiology //Pathogens. - 2019. - T. 9. - №. 1. - C. 6.
12. Martak D. et al. Comparison of pulsed-field gel electrophoresis and whole-genome-sequencing-based typing confirms the accuracy of pulsed-field gel electrophoresis for the investigation of local Pseudomonas aeruginosa outbreaks //Journal of Hospital Infection. - 2020. - T. 105. - №. 4. - C. 643-647.
13. Миронова Л. В., Афанасьев М. В., Балахонов С. В. Применение технологии пульс-электрофореза в молекулярном типировании возбудителей особо опасных инфекций //Молекулярная генетика, микробиология и вирусология. - 2015. -Т. 33. - №. 3. - С.28-32.
14. Curran В. et al. Development of a multilocus sequence typing scheme for the opportunistic pathogen Pseudomonas aeruginosa //Journal of clinical microbiology. - 2004. - T. 42. - №. 12. - C. 5644-5649.
15. Vu-Thien H. et al. Multiple-locus variable-number tandem-repeat analysis for longitudinal survey of sources of Pseudomonas aeruginosa infection in cystic fibrosis patients //Journal of clinical microbiology. - 2007. - T. 45. - №. 10. - C. 3175-3183.
16. Babenko D. et al. In silico comparison of different types of MLVA with PFGE based on Pseudomonas aeruginosa genomes //Acta Medica. - 2017. - T. 33. - C. 347.
17. Chung J. C. S. et al. Genomic variation among contemporary Pseudomonas aeruginosa isolates from chronically infected cystic fibrosis patients //Journal of bacteriology. -2012. -T. 194. -№. 18. -C. 4857-4866. -7
18. Организация работы лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности: Методические указания. МУ 1.3.2569-09. - М.: Федеральная служба по надзору в сфере защиты прав потребителей и благополучия человека, 2009. - 35 с. - 8.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing originalFreeTextLanguageCode="ru"
nonEnglishFreeTextLanguageCode="ru" dtdVersion="V1_3"
fileName="Способ дифференциации штаммов Pseudomonas aeruginosa с
помощью молекулярно-генетического типирования.xml"
softwareName="WIPO Sequence" softwareVersion="2.2.0"
productionDate="2023-04-24">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>187</ApplicationNumberText>
<FilingDate>2023-05-11</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>187</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>187</ApplicationNumberText>
<FilingDate>2023-05-24</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">Ростовский- на –Дону противочумный
институт Роспотребнадзора</ApplicantName>
<ApplicantNameLatin>Rostov-on-Don Plague Control Research Institute
of the Rospotrebnadzor</ApplicantNameLatin>
<InventorName languageCode="ru">Ковалевич Алексей
Александрович</InventorName>
<InventorNameLatin>Kovalevich Aleksey
Aleksandrovich</InventorNameLatin>
<InventionTitle languageCode="ru">Способ дифференциации штаммов
Pseudomonas aeruginosa с помощью молекулярно-генетического
типирования.</InventionTitle>
<SequenceTotalQuantity>4</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>36</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..36</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Pseudomonas aeruginosa
</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aggacatggactgcgtggcctgctgcaggcacttgg</INSDSeq_seq
uence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>40</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..40</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Pseudomonas aeruginosa
</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagtcccggatgtggtagtcgatcacgctccacaggttga</INSDSeq
_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>38</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..38</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Pseudomonas aeruginosa
</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcggcatcatcatcgagaagctgggcggtgtgtaggga</INSDSeq_s
equence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>38</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..38</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Pseudomonas
aeruginosa</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caatttcagcgccggcggcgaagacgaactggacgaac</INSDSeq_s
equence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ молекулярно-генетического типирования штаммов Klebsiella pneumoniae с использованием INDEL-маркеров | 2022 |
|
RU2796431C1 |
| Способ дифференциации штаммов Helicobacter pylori путем молекулярно-генетического типирования | 2018 |
|
RU2688434C1 |
| Способ дифференциации штаммов Legionella pneumophila путем молекулярно-генетического типирования | 2019 |
|
RU2709174C1 |
| СПОСОБ МОЛЕКУЛЯРНО-ГЕНЕТИЧЕСКОГО ВНУТРИВИДОВОГО ТИПИРОВАНИЯ V. cholerae О1 И О139 СЕРОГРУПП | 2014 |
|
RU2575046C2 |
| Способ дифференциации штаммов Francisella tularensis путем молекулярно-генетического типирования | 2020 |
|
RU2756854C1 |
| Способ генетической дифференциации штаммов Yersinia pseudotuberculosis путем молекулярно-генетического типирования | 2019 |
|
RU2736649C1 |
| Способ определения подвидов Francisella tularensis методом мультипраймерной ПЦР | 2021 |
|
RU2765495C1 |
| СПОСОБ ГЕНЕТИЧЕСКОГО INDEL-ТИПИРОВАНИЯ ШТАММОВ BRUCELLA MELITENSIS | 2019 |
|
RU2732425C1 |
| СПОСОБ МОЛЕКУЛЯРНО-ГЕНЕТИЧЕСКОГО ТИПИРОВАНИЯ ШТАММОВ САЛЬМОНЕЛЛ ПО INDEL-МАРКЕРАМ | 2022 |
|
RU2786577C1 |
| Способ дифференциации штаммов Yersinia pestis путем молекулярно-генетического типирования с использованием в качестве генетических маркеров IS-элементов | 2021 |
|
RU2767371C1 |
Изобретение относится к области медицинской микробиологии, а именно к способам молекулярно-генетического типирования штаммов Pseudomonas aeruginosa, которое может быть использовано при эпидемиологической диагностике внутрибольничных гнойно-септических, нозокомиальных инфекций на основе внутривидового типирования возбудителя. Способ дифференциации штаммов Pseudomonas aeruginosa с помощью INDEL-типирования включает выделение ДНК из исследуемого штамма P. aeruginosa, постановку ПЦР со специфическими праймерами и учет реакции с помощью электрофореза. Из ДНК исследуемого штамма выявляют четыре общих INDEL-генов, имеющих делеции определенного размера, а именно IND-РА0168, РА3324, РА0659 и РА1451, с последующей их амплификацией с помощью сконструированных специфических праймеров, выявляющих два альтернативных аллеля: к гену IND-PA0168 - праймеры AGGACATGGACTGCGTGG и CCTGCTGCAGGCACTTGG с длиной амплифицированного фрагмента 100 п. н. или 114 п. н.; к гену IND-РА3324 - праймеры GAGTCCCGGATGTGGTAGTC и GATCACGCTCCACAGGTTGA с длиной амплифицированного фрагмента 99 п. н. или 113 п. н.; к гену IND-РА0659 - праймеры GCGGCATCATCATCGAGAAG и CTGGGCGGTGTGTAGGGA с длиной амплифицированного фрагмента 106 п. н. или 112 п. н.; к гену IND-РА1451 - праймеры CAATTTCAGCGCCGGCGG и CGAAGACGAACTGGACGAAC с длиной амплифицированного фрагмента 100 п. н. или 109 п. н. Учет результатов дифференциации проводят визуально после электрофореза в 8% полиакриламидном геле в присутствии маркера молекулярных масс ДНК. Путем сравнения выявленных INDEL-генотипов между собой и с известными генотипами идентификационной таблицы устанавливают общее или различное происхождение исследуемых штаммов P. aeruginosa. Способ позволяет достоверно, быстро и с низкой себестоимостью осуществлять внутривидовую дифференциацию штаммов Р. aeruginosa. 2 з.п. ф-лы, 3 табл., 2 пр.
1. Способ дифференциации штаммов Pseudomonas aeruginosa с помощью INDEL типирования, включающий выделение ДНК из исследуемого штамма P. aeruginosa, постановку ПЦР со специфическими праймерами и учет реакции с помощью электрофореза, отличающийся тем, что из ДНК исследуемого штамма выявляют четыре общих INDEL-генов, имеющих делеции определенного размера, а именно IND-РА0168, РА3324, РА0659 и РА1451, с последующей их амплификацией с помощью сконструированных специфических праймеров, выявляющих два альтернативных аллеля:
к гену IND-PA0168 - праймеры
AGGACATGGACTGCGTGG и
CCTGCTGCAGGCACTTGG
с длиной амплифицированного фрагмента 100 п. н. или 114 п. н.;
к гену IND-РА3324 - праймеры
GAGTCCCGGATGTGGTAGTC и
GATCACGCTCCACAGGTTGA
с длиной амплифицированного фрагмента 99 п. н. или 113 п. н.;
к гену IND-РА0659 - праймеры
GCGGCATCATCATCGAGAAG и
CTGGGCGGTGTGTAGGGA
с длиной амплифицированного фрагмента 106 п. н. или 112 п. н.;
к гену IND-РА1451 - праймеры
CAATTTCAGCGCCGGCGG и
CGAAGACGAACTGGACGAAC
с длиной амплифицированного фрагмента 100 п. н. или 109 п. н.,
при этом учет результатов дифференциации проводят визуально после электрофореза в 8% полиакриламидном геле в присутствии маркера молекулярных масс ДНК, причем для каждого штамма устанавливают уникальный INDEL-генотип по четырем INDEL-генам, а путем сравнения выявленных INDEL-генотипов между собой и с известными генотипами идентификационной таблицы устанавливают общее или различное происхождение исследуемых штаммов P.aeruginosa, позволяя дифференцировать один штамм от другого.
2. Способ по п. 1, отличающийся тем, что ПЦР проводят в объеме 10 мкл и реакционная смесь содержит:
1,5 мМ MgCl, 0,2 мМ смеси дНТФ,
1,0 мкМ смеси праймеров (по 0,5 мкМ каждого праймера), 25 нг ДНК матрицы, 1 ед. ДНК-полимеразы, оставшийся объем - вода, при этом в качестве матрицы используют геномную ДНК объемом 5 мкл, которую получают из разных штаммов P.aeruginosa.
3. Способ по п. 1, отличающийся тем, что ПЦР проводят с соблюдением режимов:
1 этап - денатурация при 94°С - 10 с;
2 этап - отжиг при 60°С - 10 с;
3 этап - синтез при 72°С - 15 с (39 циклов).
| CHUNG J | |||
| C | |||
| S | |||
| et al | |||
| Genomic variation among contemporary Pseudomonas aeruginosa isolates from chronically infected cystic fibrosis patients //Journal of bacteriology | |||
| T | |||
| Кран машиниста для автоматических тормозов с сжатым воздухом | 1921 |
|
SU194A1 |
| Телеграфно-телефонная радиотрансляция | 1926 |
|
SU4857A1 |
| Способ дифференциации штаммов Helicobacter pylori путем молекулярно-генетического типирования | 2018 |
|
RU2688434C1 |
| Способ дифференциации штаммов Legionella pneumophila путем молекулярно-генетического типирования | 2019 |
|
RU2709174C1 |
| УСТРОЙСТВО ПРОЕКЦИОННОГО ДИСПЛЕЯ | 1998 |
|
RU2199144C2 |
| МИРОНОВА Л | |||
| В., АФАНАСЬЕВ М | |||
| В., БАЛАХОНОВ С | |||
| В | |||
| Применение | |||
Авторы
Даты
2024-03-26—Публикация
2023-05-24—Подача