Область техники
Настоящее изобретение относится к области метаболической инженерии и биотехнологии и может быть использовано для экспрессии интересующих генов и получения продуцентов белков и клеточных метаболитов, например, β-каротина, с помощью дрожжей Yarrowia lipolytica.
Уровень техники
Yarrowia lipolytica - уникальные дрожжи, нашедшие широкое применение в разных областях биотехнологии [1]. Промышленное использование природных изолятов дрожжей Y. lipolytica началось еще в 1950 году компанией British Petroleum (Великобритания) в качестве источника кормового белка [2]. С 2000-х годов для разработки промышленных штаммов интенсивно применяются методы генной и метаболической инженерии. На рынок выведен ряд препаратов, которые были получены с помощью рекомбинантных штаммов дрожжей Y. lipolytica. Так, например, DuPont (США) разработали препараты, обогащенные омега-3 жирными кислотами: VerlassoTM, используемый в качестве кормовой добавки для рыб, и New HarvestTM в качестве пищевой добавки [2, 3].
Дрожжи Y. lipolytica имеют статус непатогенного и безопасного микроорганизма GRAS согласно постановлению правления по санитарному надзору за качеством пищевых продуктов и медикаментов (FDA) [4]. Для данных дрожжей разработана эффективная система генно-инженерных манипуляций. Дрожжи Y. lipolytica хорошо изучены в области синтеза производных жирных кислот. В то же время дрожжи Y. lipolytica способны производить на высоком уровне различные не липидные продукты, включая поликетиды, терпены, каротиноиды, продукты, полученные из пентозофосфата и полимеры [1].
В частности, производство β-каротина, вещества с высокой биологической активностью, предшественника витамина А, описано в работе Shuliang Gaoa с соавторами [5]. Данной группой ученых проведена гетерологичная экспрессия генов, кодирующих бифункциональные ферменты фитоенсинтазу/ликопенциклазу (carRP) и фитоендегидрогеназу (carB) из Mucor circinelloides в Y. lipolytica под регуляцией конститутивных промоторов EXP1p и GPD1P, соответственно, которая позволила получить базовый рекомбинантный штамм, синтезирующий 0.3 мг/г сухого веса (4.05 мг/л) β-каротина. В работе М. Larroude с соавторами был использован конститутивный промотер TEFp для гетерологичной экспресии генов CarRP и CarB, что при экспрессии генов в одной копии и в сочетании с усилением пути синтеза внутриклеточных липидов и предшественника β-каротина геранилгеранил дифосфата позволило сконструировать штамм дрожжей Y. lipolytica, продуцирующий 8.9 мг/г сухого веса и 35.7 мг/л β-каротина [6].
Для дрожжей Y. lipolytica известны эндогенные конститутивные и индуцибельные промотеры, а также сильные квази-конститутивные гибридные промотеры [2, 7, 8, 9, 10, 11, 12, 13]. Промоторы позволяют осуществлять регуляцию и экспрессию генов на уровне транскрипции и выбор промотора имеет важное значение при конструировании трансформантов с высоким титром целевого продукта.
Технической проблемой, на решение которой направлено представленное изобретение является расширение арсенала конститутивных промоторов для генной и метаболической инженерии дрожжей Y. lipolytica, а также расширение арсенала трансформантов дрожжей Y. lipolytica, продуцирующих β-каротин.
Раскрытие сущности изобретения
Техническим результатом является получение трансформанта дрожжей Yarrowia lipolytica, продуцирующего β-каротин с участием нуклеотидной последовательности, обладающей активностью промотора.
Поставленная задача решена тем, что предложено применение фрагмента ДНК, включающего нуклеотидную последовательность SEQ ID NO: 1 или последовательность, обладающую, по меньшей мере, 80% гомологией к ней и идентичной функциональной активностью, в качестве промотора в дрожжах Yarrowia lipolytica.
Поставленная задача решена тем, что предложен трансформант дрожжей Yarrowia lipolytica, продуцирующий β-каротин, содержащий в составе хромосомы, по крайней мере, одну копию нуклеотидной последовательности SEQ ID NO: 1 или последовательности, обладающей, по меньшей мере, 80% гомологией к ней и проявляющей промоторную активность, а также, по крайней мере, по одной копии каждого из следующих оптимизированных синтетических генов,
- CarRP M. circinelloides, нуклеотидная последовательность которого приведена в перечне последовательностей под номером SEQ ID NO: 2, кодирующий бифункциональный фермент фитоен синтазу/ликопин β-циклазу,
- GGPPs7 Synechococcus sp. нуклеотидная последовательность которого приведена в перечне последовательностей под номером SEQ ID NO: 3, кодирующий геранилгеранил пирофосфат синтазу,
- CarB M. circinelloides, последовательность которого приведена в перечне последовательностей под номером SEQ ID NO:4, кодирующий фитоендегидрогеназу.
Достижение технического результата основано на установленном нами факте, что последовательность ДНК, приведенная в перечне последовательностей под номером SEQ ID NO: 1, расположенная перед геном дрожжей Y. lipolytica AraDH2 [14], проявляет промоторную активность в дрожжах Y. lipolytica по отношению к ферментам, ответственным за синтез каротиноидов. С этой целью возможно также использование последовательности, обладающей, по меньшей мере, 80% гомологией к последовательности SEQ ID NO: 1 и указанной (промоторной) активностью.
Здесь термин "гомология" относится к степени идентичности с заданной нуклеотидной последовательностью и может быть определена с использованием любого алгоритма, известною в данной области техники, например, BLAST [15] или FASTA, введенного Pearson [16]. На основе алгоритма BLAST были разработаны программы, известные как BLASTN или BLASTX [17].
Очевидно, что любой полинуклеотид, имеющий полинуклеотидную последовательность с гомологией, описанной выше, и включающий делецию, модификацию, замену или добавление одной или нескольких последовательностей, входит в объем настоящего изобретения, при условии что этот полинуклеотид сохраняет биологические активности, по существу идентичные или соответствующие активностям нуклеотидной последовательности SEQ ID NO: 1.
Конструирование экспрессионных кассет при получении трансформанта дрожжей осуществляют стандартными методами генетической инженерии [18] с использованием генетических элементов, подходящих для работы с дрожжами рода Yarrowia.
В качестве терминаторов транскрипции могут быть использованы Aco3, CYC1, Guo, LYP1, LYP2, PEX3, PEX16, PEX20, PHO5, TEF1, ScADH1, ScPGK1, ScENO2, Tsynth2, Tsynth7, Tsynth8, Tsynth10, Tsynth22, Tsynth27, Tsynth30, XPR2 [7, 19, 20, 21, 22].
Введение генов может быть осуществлено как хромосомальной, так и эписомальной интеграцией, в качестве сайтов для гомологичной интеграции могут быть использованы последовательности, гомологичные участкам хромосомы дрожжей рода Yarrowia. В целях повышения эффективности экспрессии мРНК с интегрированных последовательностей при трансляции предпочтительно использовать гетерологичные гены, кодон-оптимизированные для дрожжей Y. lipolytica.
Трансформацию экспрессионной кассеты в клетки дрожжей Y. lipolytica осуществляют любым подходящим способом, в частности, методом электоропорации [23] или методом с использованием ацетата лития [24].
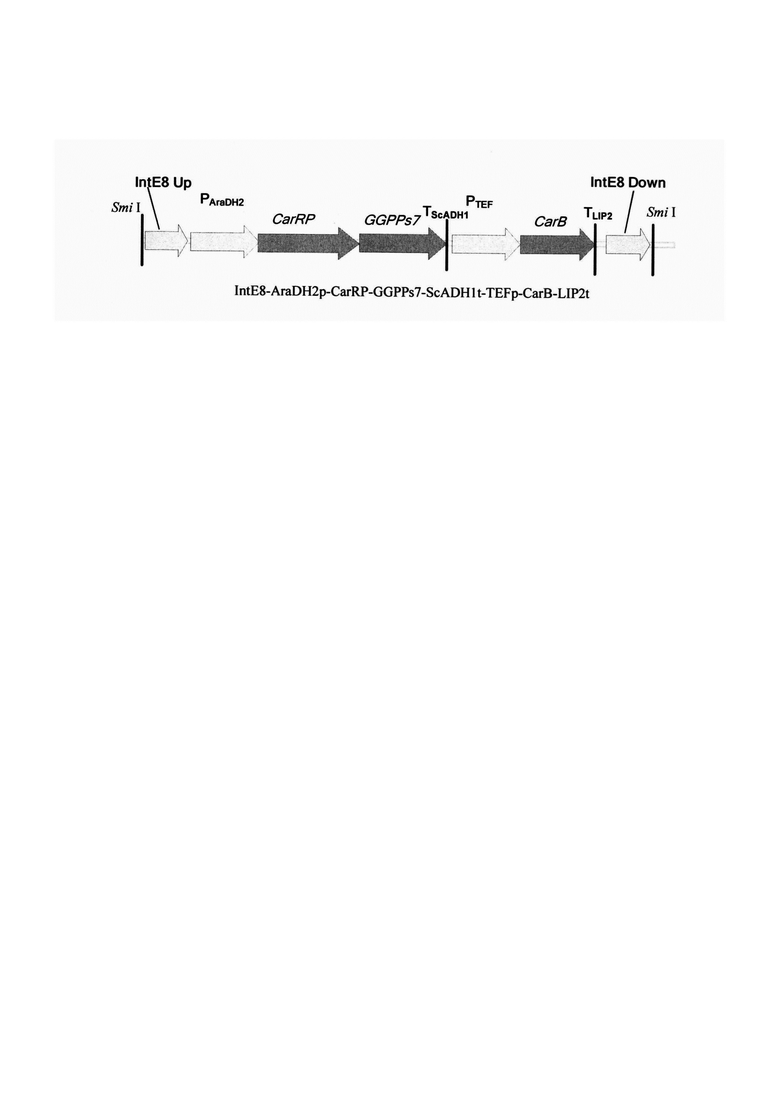
Краткое описание чертежей
На фигуре показана экспрессионная кассета IntE8-AraDH2p-CarRP-GGPPs7-ScADH1t-TEFp-CarB-LIP2t.
Осуществление изобретения
Пример 1: Получение последовательности SEQ ID NO: 1
Последовательность SEQ ID NO: 1 получают следующим образом.
Последовательность перед геном AraDH2 амплифицируют с геномной ДНК штамма Y. lipolytica W29 (ВКПМ Y-3178) с помощью пар праймеров:
TTTCACGCCCCGATCAAGC (AraDH2-F)
GATTGATTTTGTGTGTGTTGTG (AraDH2-R).
Полученный фрагмент ДНК очищают после электрофореза в 1% агарозном геле и секвинируют по праймерам AraDH2-F/AraDH2-R. Нуклеотидная последовательность AraDH2p полученного фрагмента приведена в перечне последовательностей под номером SEQ ID NO: 1.
Пример 2: Получение трансформанта, продуцирующего β-каротин
В качестве штамма-реципиента используют штамм линии W29 Y. lipolytica ВКПМ Y-4967.
Получают плазмиду pE8UA2.2-AraDH2p-CarRP-GGPPs7-ScADH1t-TEFp-CarB-LIP2t, которую собирают методом Golden Gate [22] из следующих плазмид:
1. Плазмида pE8US1.1-AraDH2p-CarRP-GGPPs7-ScADH1t
1.1 Плазмида pYTK001-AraDH2
На основе последовательности SEQ ID NO: 1 получают фрагмент AraDH2-GGA с помощью амплифицикации по паре праймеров:
ATATCGTCTCATCGGGGTCTCAACGGATCGATTTTCACGCCCCGATCAAGC (AraDH2-GGA-F)
TTAACGTCTCAGGTCGGTCTCACATTGGATCCGATTGATTTTGTGTGTGTTGTGG (AraDH2-GGA-R).
Фрагмент AraDH2-GGA собирают с плазмидой №175709 (Addgene) в единую плазмиду pYTK001-AraDH2 с использованием эндонуклеазы рестрикции Esp3I по методу Golden Gate
1.2 Плазмида pYTK001-CarRP-GGPPs7
Плазмиду pYTK001-CarRP-GGPPs7 конструируют на базе вспомогательной синтезированной плазмиды №175709 (Addgene) и синтезированных фрагментов CarRP-fus (ген CarRP приведен в перечне последовательностей под номером SEQ ID NO: 2) и fus-GGPPs7 (ген GCPPs7 приведен в перечне последовательностей под номером SEQ ID NO: 3).
Здесь и далее все гетерологичные гены кодон-оптимизируют для Y. lipolytica с помощью онлайн программы GenSmart™ (https://www.genscript.com/gensmart-free-gene-codon-optimization.html) и синтезируют в виде фрагментов ДНК (синтез осуществлен компанией «Twist Bioscience Corporation» (США).
Фрагмент CarRP-fus амплифицируют с синтезированной последовательности гена CarRP (SEQ ID NO: 2) с помощью пар праймеров:
ATATCGTCTCATCGGGGTCTCAAATGCTCCTGACCTACATGGA (GGA-CarRP-F)
TTATACGTCTCTGGTCGGTCTCAGGCAATAGGGGCGGTGTAGAGATT (GGA-CarRP-R).
Фрагмент fus-GGPPs7 - амплифицируют с синтезированной последовательности гена GGPPs7 (SEQ ID NO: 3) с помощью пар праймеров:
ATATCGTCTCATCGGGGTCTCATGCCACGTCTAGCACAACCCACTGTAGACAAGGAAAGATGCGGAATCTCAACACCATCGGAGGCGGATCCGTGGCTCAAACTTTTAACCTG (GGA-GGPPs7-F)
TATACGTCTCTGGTCGGTCTCATAGATTAATGCTGTCGCCGGGTG (GGA-GGPPs7-R).
Полученные фрагменты очищают после электрофореза в 1% агарозном геле.
ПЦР-фрагменты CarRP-fus и fus-GGPPs7 собирают с плазмидой №175709 (Addgene) в единые плазмиды pYTK001-CarRP-fus, pYTK001-fus-GGPPs7, соответственно, с использованием эндонуклеазы рестрикции Esp3I по методу Golden Gate.
ДНК последовательность CarRP-fus на полученной плазмиде pYTK001-CarRP-fus секвенируют с помощью праймеров:
TTTTGCTCACATGTTCTTTCCT (pYT-seqF)
TAATGACCTCAGAACTCCATC (pYT-seqR)
ATCAGAGCAAAGAACATGAACT (CarRP-GGA-seq-1-R)
TATTCCTCTGCTGGGTCGAT (CarRP-GGA-seq-2-F)
TGTCACGTGCAATGTTGGTG (CarRP-GGA-seq-3-R).
ДНК последовательность fus-GGPPs7 на полученной плазмиде pYTK001-fus-GGPPs7 секвенируют с помощью праймеров:
TTTTGCTCACATGTTCTTTCCT (pYT-seqF)
TAATGACCTCAGAACTCCATC (pYT-seqR)
TGGCCTTGCCTTCCGATTC (GGPPs7-GGA-seq-R).
Из полученных плазмид: №175741 (Addgene), pYTK001-AraDH2, pYTK001-CarRP-fus, pYTK001-fus-GGPPs7, №175724 (Addgene) с использованием эндонуклеазы рестрикции Eco31I конструируют единую экспрессионную плазмиду pE8US1.1-AraDH2p-CarRP-GGPPs7-ScADH1t.
2. Плазмида pE8US 1.2-TEFp-CarB-LIP2t
Экспрессионную плазмиду pYTK001-CarB конструируют на базе вспомогательной синтезированной плазмиды №175709 (Addgene) и синтезированного фрагмента CarB (ген CarB приведен в перечне последовательностей под номером SEQ ID NO: 4). Фрагмент CarB амплифицируют с последовательности SEQ ID NO: 4 с помощью пар праймеров GGA-fr-solo-F и GGA-fr-solo-R.
ATATCGTCTCATCGGGGTCT (GGA-fr-solo-F)
TATACGTCTCTGGTCGGTCT (GGA-fr-solo-R).
Полученный фрагмент очищают после электрофореза в 1% агарозном геле.
ПЦР-фрагмент CarB собирают с плазмидой №175709 (Addgene) в единую плазмиду pYTK001-CarB с использованием эндонуклеазы рестрикции Esp3I по методу Golden Gate. ДНК последовательность CarB на полученной плазмиде секвенируют с помощью праймеров:
TTTTGCTCACATGTTCTTTCCT (pYT-seqF)
TAATGACCTCAGAACTCCATC (pYT-seqR)
TTCTGCTTGGCAATGGCTTC (CarB-GGA-seq1-R)
TCTACTCGTTGCTGCAGTAC (CarB-GGA-seq1-F)
AAAGACTTGACCACCTGGTC (CarB-GGA-seq2-R).
Единую экспрессионную плазмиду pE8US1.2-TEFp-CarB-LIP2t конструируют из следующих плазмид: №175742 (Addgene), №175721 (Addgene), pYTK001-CarB, №175723 (Addgene) с использованием эндонуклеазы рестрикции Eco31I.
3. Плазмида pE8UA2.2-AraDH2p-CarRP-GGPPs7-ScADH1t-TEFp-CarB-LIP2t
Плазмиду pE8UA2.2-AraDH2p-CarRP-GGPPs7-ScADH1t-TEFp-CarB-LIP2t собирают из трех плазмид: №175700 (Addgene), pE8US1.1-AraDH2p-CarRP-GGPPs7-ScADH1t и pE8US1.2-TEFp-CarB-LIP2t с использованием эндонуклеазы рестрикции Esp3I.
Фрагмент ДНК IntE8-AraDH2p-CarRP-GGPPs7-ScADH1t-TEFp-CarB-LIP2t (чертеж) получают путем обработки плазмиды pE8UA2.2-AraDH2p-CarRP-GGPPs7-ScADH1t-TEFp-CarB-LIP2t эндонуклеазой рестрикции и используют совместно со вспомогательной кольцевой плазмидой №175704 (Addgene) для ко-трансформации штамма Y. lipolytica ВКПМ Y-4967.
Трансформанты отбирают на среде YPSuc Nat (мас. %: дрожжевой экстракт - 1, пептон - 2, сахароза - 1, агар - 1, вода - остальное) по устойчивости к антибиотику нурсеотрицину. У трансформантов изолируют геномную ДНК с помошью Yeast DNA Extraction Kit (Thermo Scientific, США), и проводят ПЦР-анализ на правильность интеграции генетической кассеты с помощью праймеров:
TAATCATACTGCGCCGTGCC (IntE8-chr-F)
GGCTCTTAAAAGCGTTCAGA (IntE8-chr-R).
Полученный трансформант I характеризуется наличием генетической кассеты IntE8-AraDH2p-CarRP-GGPPs7-ScADH1t-TEFp-CarB-LIP2t, интегрированной в локус IntE8 на хромосоме E дрожжей Y. lipolytica.
Отобранный трансформант депонирован в Биоресурсном Центре Всероссийская Коллекция Промышленных Микроорганизмов (БРЦ ВКПМ) НИЦ «Курчатовский институт» (117545 Москва, 1-ый Дорожный пр-д, д.1) как штамм дрожжей Y. lipolytica ВКПМ Y-5032.
Признаки штамма дрожжей Y. lipolytica ВКПМ Y-5032
Культурально-морфологические признаки
Суточная культура в жидкой YPD среде (мас. %: дрожжевой экстракт - 1, пептон - 2, глюкоза - 1, вода - остальное) представлена овальными, удлиненно-овальными, округлыми клетками размером 4,6-6,0×4,5-12,5 мкм. Почкование полярное или латеральное, на узком основании. К третьим суткам большинство клеток почкуются и образуют истинный и псевдомицелий.
При росте в жидкой среде YPD при 28°С в течение 24 ч культивирования - жидкость мутная, осадок оранжевый, коагуляции не наблюдается, пристеночных пленок не образует.
Клетки хорошо растут на простых питательных средах. Колонии на мальтоагаре (возраст 1 неделя) оранжевого цвета, пастообразные, слегка приподнятые в центре, морщинистые, с фестончатым краем. Штрих на мальтоагаре непрерывный, плоский, блестящий, оранжевого цвета, пастообразный, края ровные, со временем становится складчатым. Спор не образует.
Физиолого-биохимические признаки.
Облигатный аэроб. Сахара не сбраживает. Ассимилирует: сахарозу, глюкозу, D-галактозу (медленно), L-сорбозу, D-рибозу, этанол, глицерин, эритрит, адонит, D-маннит, сорбит и молочную, янтарную, лимонную, глюконовую кислоты. Не ассимилирует: мальтозу, лактозу, целлобиозу, трегалозу, мелибиозу, раффинозу, мелицитозу, инулин, крахмал, D-ксилозу, L- и D-арабинозу, раммозу, дульцит, инозит, D-глюкозамин и глюкуроновую, 2-кетоглюконовую, 5-кетоглюконовую кислоты. Не ассимилирует нитраты, жирные кислоты и алканы. Не растет в безвитаминной среде, требует присутствия в среде тиамина, не требует биотина.
Оптимальное значение рН для роста 5,5-7,0. Не растет при 37°С. Максимальная температура роста 35°С. Разжижает желатин. Гидролизует мочевину.
Пример 3: Культивирование штамма Y. lipolytica ВКПМ Y-5032 в пробирках
Отобранный штамм Y. lipolytica ВКПМ Y-5032 культивируют при 30°С и постоянном перемешивании (250 об/мин) в течение 5 суток в пробирках (50 мл) с рабочим объемом 10 мл в жидкой среде YP (мас. %: дрожжевой экстракт - 1, пептон - 1, вода - остальное) с добавлением разных источников углерода: сахарозы 9 мас. %, или глицерина 9 мас. %, или глюкозы 9 мас. %. Результаты культивирования приведены в табл. 1.
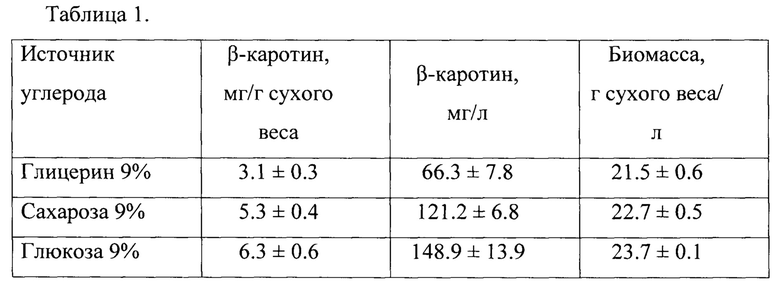
Полученные результаты показывают, что использование последовательности SEQ ID NO: 1 в качестве промотора для экспрессии генов пути синтеза β-каротина, позволяет получить трансформант, хорошо растущий на средах с разными источниками углерода и позволяющий получить продукцию β-каротина с более высоким титром (до 148.9 мг/л) по сравнению с таковым при использовании промоторов EXP1p и GPD1p [5] и TEFp [6] (4.05 мг/л и 35,7 мг/л, соответственно).
1. Miller K.K., Alper H.S. (2019). Appl Microbiol Biotechnol, 103(23-24), 9251-9262. doi:10.1007/s00253-019-10200-х
2. Madzak С. (2015). Appl Microbiol Biotechnol, 99(11), 4559-4577. doi:10.1007/s00253-015-6624-z
3. Xue Z., Sharpe P.L. et al. (2013). Nat Biotechnol, 31(8), 734-740. doi:10.1038/nbt.2622
4. https://www.ncbi.nlm.nih.gov/pmc/articies/PMC8307478/
5. Gao S., Tong Y. et al. (2017). Metab Eng, 41, 192-201. doi: 10.1016/j.ymben.2017.04.004
6. Larroude, M., Celinska et al. (2018). Biotechnol Bioeng, 115(2), 464-472. doi:10.1002/bit.26473
7. Darvishi F., Ariana M. et al. (2018). Appl Microbiol Biotechnol, 102(14), 5925-5938. doi:10.1007/s00253-018-9099-x
8. Park Y.K., Korpys P. et al. (2019). FEMS Yeast Res, 19(1). doi: 10.1093/femsyr/foy 105
9. Kamineni A., Chen S. et al. (2020).10 FEMS Yeast Res, 20(5). doi:10.1093/femsyr/foaa035
10. US 20060115881
11. Madzak C., Treton B. et al. (2000). J Mol Microbiol Biotechnol, 2(2), 207-216.
12. Tai M., Stephanopoulos G. (2013). Metab Eng, 15, 1-9. doi:10.1016/j.ymben.2012.08.007
13. Sun M. L., Shi T. Q. et al (2022). Bioresour Technol, 347, 126717. doi:10.1016/j.biortech.2022.126717
14. Wang N., Chi P. et al (2020). Biotechnol Biofuels, 13, 176. doi:10.1186/s13068-020-01815-8
15. Karlin and Altschul, Pro. Natl. Acad. Sci. USA, 90, 5873, 1993
16. Methods Enzymol., 183, 63, 1990
17. http://www.ncbi.nlm.nih.gov
18. Sambrook J., Maniatis T. et al (1989). Molecular cloning: laboratory Mannual. 2nd ed. New York, USA: Cold Spring Harbor Laboratory Press.
19. Curran K.A., Morse N. J. et al (2015). ACS Synth Biol, 4(7), 824-832. doi:10.1021/sb5003357
20. Larroude M., Park Y. K. et al (2019). Microb Biotechnol, 12(6), 1249-1259. doi:10.1111/1751-7915.13427
21. EP 2649887 B1
22. https://www.researchsquare.com/article/rs-1570357/v1
23. Saraya R., Gidijala L. et al (2014). Metods Mol Biol, 1152, 43-62. doi:10.1007/978-1-4939-0563-8_3
24. Berardi E., Thomas D. Y. (1990) Current Genetics 18, 169-170. doi:10.1007/BF00312606.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing originalFreeTextLanguageCode="ru"
nonEnglishFreeTextLanguageCode="ru" dtdVersion="V1_3"
fileName="Promotor.xml" softwareName="WIPO Sequence"
softwareVersion="2.1.2" productionDate="2022-09-20">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>PCT/RU2022/009999</ApplicationNumberText>
<FilingDate></FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>AB134</ApplicantFileReference>
<ApplicantName languageCode="ru">Национальный исследовательский
центр «Курчатовский институт»</ApplicantName>
<ApplicantNameLatin>National Research Center Kurchatov
Institute</ApplicantNameLatin>
<InventionTitle languageCode="ru">Промотор для экспрессии генов и
его использование при конструировании трансформанта дрожжей Yarrowia
lipolytica, продуцирующего β-каротин</InventionTitle>
<SequenceTotalQuantity>4</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>576</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..576</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Yarrowia lipolytica</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tttcacgccccgatcaagcacttgttccacatgtttctatactctgcgt
ctgcgcagacgtcattttgcatgtctgaagtgggtttggggtttgtatggctgtcgagtaaagagcgcat
ggagatttttgtaaaattctagaaaaaatacttgtagtttcctcgctcatcatagttatcatcctggact
ctcacgttccacccccccacgctcactactagcacttgctgtagtcccctctctcgtctatctaccgccc
taatgccgactgcactatgcgatgtccagataaggtagcaatccacacttctaggtttgtgaggtggtgt
tgaggtaccgacttgattcagacgactgaaaggacctccagctacatctaaaatcccagaggacgtgcga
agaaggtctttaagcccctttgtgttctagcgaccaaacccaacccgtcaaggcgacatacatcacgtcg
acctcgtcccattgctgaacctaaacccacgcactccttgtataacaggacgtggtcaactcaactctct
cttgtacacatacaccacaacacacacaaaatcaatc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>1842</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..1842</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Yarrowia lipolytica</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgctcctgacctacatggaggtgcatctgtactacactttgcctgttc
tgggagtcctgtcttggctgtctcgaccctactacacggccacagacgccctcaaattcaagtttctgac
tctagttgcattcaccactgcttccgcttgggacaactacattgtctaccacaaggcctggtcatactgc
cctacctgtgtgactgctgtcattggttacgtgccgttggaagagtacatgtttttcattatcatgactc
tgctaaccgtggcctttactaatctagtgatgagatggcacctccactctttctttatccgaccagagac
tcccgtcatgcagtccgtccttgtgcgacttgtccccatcacagcacttctcatcaccgcctacaaggcg
tggcatctcgcagttcctggaaaaccgctgttctatggaagttgtatcttgtggtacgcatgccccgtgc
tggccctgctgtggttcggagccggcgagtacatgatgcgacgaccattggctgtccttgtctcgattgc
gctcccgacgttattcctctgctgggtcgatgtcgtggctattggcgctggaacatgggacatcagcctg
gccacatctacaggtaagtttgtcgtgcctcatttacccgttgaggagttcatgttctttgctctgatca
acactgtcttggtgtttggtacctgtgctattgatcgcaccatggcaatcctacatcttttcaagaacaa
atctccttaccaacgtccttaccagcattcgaaatcattcctgcaccagattcttgaaatgacctgggcc
ttctgtttgccagaccaagtcctccacagcgacaccttccacgacctgtccgtgtcctgggacatcctgc
gcaaggcttcaaagagtttttacacggcatctgctgtgttccccggtgacgtgcgacaggagctcggcgt
gctctatgccttttgtcgagccaccgacgacctgtgtgataacgagcaggtccccgtccagactcgaaag
gaacagctgattctgacacatcagttcgtgtctgatctgtttggacaaaagacttccgctcccactgcga
ttgactgggatttctacaacgaccagctccccgccagttgcatttctgcgtttaaatcctttactcgtct
gaggcatgttctcgaggccggtgccataaaggagctgctcgatggctacaagtgggatctcgaacgaaga
tcgatccgggatcaggaggatcttcggtactattctgcctgtgttgcgtcgtccgttggtgagatgtgca
cgcggataatcctggctcacgccgataagcccgcctccagacagcagacccagtggatcatccaacgggc
ccgagagatgggcctggtactccagtacaccaacattgcacgtgacattgtcacggactcggaggaactc
ggacgttgctacctgccccaggactggctcactgagaaggaagttgctttgatccagggcgggttggctc
gagaaattggagaggagagattgctttcgctcagccacagactcatctaccaggctgacgagctaatggt
tgttgccaacaagggtatcgacaagctgccctcccactgccagggcggcgtacgcgctgcctgcaacgtg
tatgcctccatcggaaccaagctcaagtcctataagcaccactacccgtcgcgagcgcacgtggggaaca
gcaagcgagtggagattgctctgctttccgtgtataatctctacaccgcccctattgccacgtctagcac
aacccactgtagacaaggaaagatgcggaatctcaacaccatc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>894</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..894</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q3">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Yarrowia lipolytica</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atggtggctcaaacttttaacctggatacctacctatctcagcgacagc
agcaagtcgaggaggcattgtcagctgctcttgttccggcgtatcctgagcgaatctacgaggctatgcg
atactctttgctcgctggcggcaaacgtctgcgacccatcctctgtctggcagcctgcgagcttgctgga
gggagtgtggaacaggccatgcccactgcttgtgctcttgagatgatccacaccatgtctctgattcacg
acgatcttcccgccatggacaatgatgacttcagaagaggtaaaccaaccaaccacaaggtgtttggcga
ggacatcgccatcttggcaggcgacgccctcttagcctatgcctttgagcacattgcctcgcagacgcgc
ggagtccctccccagctggtgctgcaggttattgctcgaattggtcatgctgttgccgccaccggtcttg
tcggaggccaggtggtagatctggaatcggaaggcaaggccatctccctagagactctggagtacattca
ttcccataagacgggggcgttgttggaggcgtccgtcgtgtctggaggtattctcgcaggagccgacgag
gagctcctcgctcggctcagccactacgcccgagatataggtctggccttccagattgtcgacgacattc
tggacgtgacagccacttctgagcaactcggaaagaccgcaggcaaggaccaggctgcagcgaaggccac
atacccgtcgctgctgggcctggaagcctcacgtcagaaggcggaggaactcatccagtccgccaaggag
gctctgcgaccctacggcagccaagccgaacctctgctcgccctggctgatttcatcacccggcgacagc
attaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>1789</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..1789</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Yarrowia lipolytica</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atatcgtctcatcggggtctcaaatgtccaaaaagcacattgtgatcat
tggcgcaggagttggaggtactgccaccgcggcaagactggctcgagaggggtttaaggtcacggttgtt
gagaagaacgactttggaggaggccgatgcagccttatccaccaccagggtcatcgattcgatcaaggcc
ccagcctgtacctcatgcctaagtactttgaggacgcatttgctgatctcgacgagcgtattcaggacca
tctggagttgctcagatgtgacaacaactacaaggtgcactttgacgacggcgagtcgatccagctctct
agcgacctaacccgaatgaaggcagaactggatcgagtggagggtcctctggggttcggccggttcttgg
atttcatgaaggagactcacatccactacgagtctggtacgctgatcgctctcaaaaaaaactttgaatc
aatttgggacctgattcgaatcaagtatgcccccgaaatcttccggcttcatctttttggaaagatttac
gaccgcgcctctaagtacttcaagaccaagaaaatgcgcatggcttttaccttccagaccatgtacatgg
gcatgtcaccttatgacgctcccgccgtctactcgttgctgcagtacaccgagtttgccgagggaatctg
gtacccacgaggcggcttcaacatggtggttcaaaagcttgaagccattgccaagcagaaatatgatgct
gagttcatctacaacgcacccgtggccaaaatcaacaccgacgatgccacaaaacaggtgaccggtgtca
ccttagaaaatggtcatataatcgatgcagatgctgtcgtatgcaacgccgatctcgtgtatgcttacca
caacctgttacctccttgtcgatggacccagaacacgttggcctccaagaagctgacttcttccagcatc
tctttctactggtccatgtcgactaaggttcctcaacttgacgtgcataacatctttcttgccgaggcct
accaggagagtttcgacgaaattttcaaggacttcggcttgccctccgaggcttcgttctatgtgaatgt
gccctcgaggattgacccctctgctgcaccagacggcaaggattctgtgattgtactcgtccccattgga
cacatgaagtccaagacaggcgacgcctccactgaaaactaccccgccatggtcgacaaggcccgaaaga
tggttctcgccgtcattgaacgtagactgggtatgtctaacttcgccgacctcattgagcacgagcaggt
caacgatcctgctgtgtggcagagtaagttcaacctctggagaggatcgattctgggactctcgcacgac
gtcctgcaagtgctgtggtttcgtccttcaaccaaggactcgacaggtcgatacgataatctcttctttg
tcggtgcctcaacgcatcccggaactggtgtaccaatagttcttgcgggctccaaacttacatccgacca
ggtggtcaagtcttttggtaagactccgaagccccggaagatcgagatggagaacacacaggccccgctg
gaggagcctgatgcggagtccacttttcccgtttggttctggctgcgggctgccttctgggtcatgttta
tgttcttctactttttcccccagtctaacggccagactcctgcgtctttcattaacaatctgctaccgga
ggtgttccgagtccacaactccaatgttatctaatctatgagaccgaccagagacgtata</INSDSeq_
sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Трансформант дрожжей Yarrowia lipolytica, продуцирующий кантаксантин | 2023 |
|
RU2827562C1 |
| Модифицированная нуклеотидная последовательность, кодирующая полипептид, обладающий активностью фитоен синтазы и геранилгеранилпирофосфат синтазы (варианты), ее использование при конструировании трансформантов дрожжей Yarrowia lipolytica, продуцирующих каротиноиды | 2021 |
|
RU2794980C1 |
| Трансформант дрожжей Yarrowia lipolytica, продуцирующий линалоол | 2022 |
|
RU2809554C1 |
| Штамм дрожжей Yarrowia lipolytica - продуцент линалоола | 2023 |
|
RU2819537C1 |
| СПОСОБ ПОЛУЧЕНИЯ ЯНТАРНОЙ КИСЛОТЫ С ИСПОЛЬЗОВАНИЕМ ДРОЖЖЕЙ, ПРИНАДЛЕЖАЩИХ К РОДУ Yarrowia | 2009 |
|
RU2422526C2 |
| ИНТЕГРАТИВНАЯ ГЕНЕТИЧЕСКАЯ КОНСТРУКЦИЯ pQ-SRUS ДЛЯ ПОЛУЧЕНИЯ ШТАММОВ ДРОЖЖЕЙ Yarrowia lipolytica, ОБЛАДАЮЩИХ СПОСОБНОСТЬЮ К ГОМОЛОГИЧНОЙ РЕКОМБИНАЦИИ ГЕНОМА МИТОХОНДРИЙ ЗА СЧЁТ ЭКСПРЕССИИ ГЕНА БЕЛКА RecA БАКТЕРИАЛЬНОГО ПРОИСХОЖДЕНИЯ | 2014 |
|
RU2562869C1 |
| ГЕНЕТИЧЕСКАЯ КОНСТРУКЦИЯ ДЛЯ ЭКСПОЗИЦИИ БЕЛКА НА ПОВЕРХНОСТИ КЛЕТОЧНОЙ СТЕНКИ ДРОЖЖЕЙ Yarrowia lipolytica | 2010 |
|
RU2451749C1 |
| МУЛЬТИЗИМЫ И ИХ ИСПОЛЬЗОВАНИЕ В ПОЛУЧЕНИИ ПОЛИНЕНАСЫЩЕННЫХ ЖИРНЫХ КИСЛОТ | 2008 |
|
RU2517608C2 |
| ДРОЖЖИ РОДА YARROWIA, ОБЛАДАЮЩИЕ СПОСОБНОСТЬЮ ВНУТРИКЛЕТОЧНО НАКАПЛИВАТЬ СЛОЖНЫЕ ЭФИРЫ ЖИРНЫХ КИСЛОТ, И СПОСОБ МИКРОБИОЛОГИЧЕСКОГО СИНТЕЗА ТАКИХ ЭФИРОВ | 2013 |
|
RU2539744C1 |
| Трансформант дрожжей Ogataea haglerorum, продуцирующий бета-маннаназу, содержащий в составе хромосомы синтетический ген MANS | 2020 |
|
RU2764793C1 |
Изобретение относится к биотехнологии. Предложено применение фрагмента ДНК, включающего нуклеотидную последовательность SEQ ID NO: 1 или последовательность, обладающую по меньшей мере 80% гомологией к ней и идентичной функциональной активностью, в качестве промотора в дрожжах Yarrowia lipolytica. Также предложен трансформант дрожжей Yarrowia lipolytica, продуцирующий β-каротин и содержащий в составе хромосомы по крайней мере одну копию нуклеотидной последовательности SEQ ID NO: 1 или последовательности, обладающей по меньшей мере 80% гомологией к ней и проявляющей промоторную активность, а также по крайней мере по одной копии каждого из следующих оптимизированных синтетических генов: CarRP М. circinelloides, нуклеотидная последовательность которого приведена в перечне последовательностей под номером SEQ ID NO: 2, кодирующий бифункциональный фермент фитоен синтазу/ликопин Р-циклазу, GGPPs7 Synechococcus sp., нуклеотидная последовательность которого приведена в перечне последовательностей под номером SEQ ID NO: 3, кодирующий геранилгеранил пирофосфат синтазу, CarB Mucor circinelloides, нуклеотидная последовательность которого приведена в перечне последовательностей под номером SEQ ID NO: 4, кодирующий фитоендегидрогеназу. Также предложен рекомбинантный штамм дрожжей Yarrowia lipolytica ВКПМ Y-5032, являющийся продуцентом β-каротина. Изобретение обеспечивает эффективное получение трансформанта Yarrowia lipolytica, продуцирующего β-каротин. 3 н.п. ф-лы, 1 ил., 1 табл., 3 пр.
1. Применение фрагмента ДНК, включающего нуклеотидную последовательность SEQ ID NO: 1 или последовательность, обладающую по меньшей мере 80% гомологией к ней и идентичной функциональной активностью, в качестве промотора в дрожжах Yarrowia lipolytica.
2. Трансформант дрожжей Yarrowia lipolytica, продуцирующий β-каротин, содержащий в составе хромосомы по крайней мере одну копию нуклеотидной последовательности SEQ ID NO: 1 или последовательности, обладающей по меньшей мере 80% гомологией к ней и проявляющей промоторную активность, а также по крайней мере по одной копии каждого из следующих оптимизированных синтетических генов, CarRP М. circinelloides, нуклеотидная последовательность которого приведена в перечне последовательностей под номером SEQ ID NO: 2, кодирующий бифункциональный фермент фитоен синтазу/ликопин Р-циклазу, GGPPs7 Synechococcus sp., нуклеотидная последовательность которого приведена в перечне последовательностей под номером SEQ ID NO: 3, кодирующий геранилгеранил пирофосфат синтазу, CarB Mucor circinelloides, нуклеотидная последовательность которого приведена в перечне последовательностей под номером SEQ ID NO: 4, кодирующий фитоендегидрогеназу.
3. Рекомбинантный штамм дрожжей Yarrowia lipolytica ВКПМ Y-5032 - продуцент β-каротина.
| Паровая форсунка | 1929 |
|
SU17557A1 |
| Способ размножения копий рисунков, текста и т.п. | 1921 |
|
SU89A1 |
| Паровая форсунка | 1929 |
|
SU17557A1 |
| LARROUDE M | |||
| et al | |||
| A synthetic biology approach to transform Yarrowia | |||
Авторы
Даты
2024-05-21—Публикация
2022-11-25—Подача