Изобретение относится к области биотехнологии и может быть использовано для получения каротиноида кантаксантина.
Уровень техники
Каротиноид кантаксантин - краситель с высокими антиоксидантными свойствами, является ценным продуктом, имеющим множество применений на рынке, в том числе в качестве кормовой добавки для животных, особенно для кормления домашней птицы. Кантаксантин является субстратом для производства другого кетокаротиноида, представляющего коммерческий интерес - астаксантина [Rebelo В. A., Farrona S., et all. (2020). Plants (Basel), 9(8). doi:10.3390/plants9081039]. Объем рынка кантаксантина оценивается в 75 миллионов долларов США.
Кантаксантин получают в основном химическим синтезом, который, однако, не отвечает требованиям биобезопасности [Chen М, Li М., et al. (2022). ACS Synth Biol, 11(1), 325-333. doi:10.1021/acssynbio.lc00437]. Биотехнологическое производство с использованием клеточных фабрик представляет собой многообещающий альтернативный путь получения кантаксантина.
Известен [Chen М., Li М., et al. (2022). ACS Synth Biol, 11(1), 325-333. doi:10.1021/acssynbio.lc00437] рекомбинантный штамм дрожжей Saccharomyces cerevisiae, способный накапливать кантаксантин, несущий укороченный вариант гена tHMGl S. cerevisiae, кодирующего 3-гидрокси-3-метилглютарил-кофермент А редуктазу, мутантный вариант гена crtE и гены crtI и CrtYB Xanthophyllomyces dendrorhous, кодирующие геранилгеранилдифосфат синтазу, фитоендесатуразу, бифункциональную ликопинциклазу/фитоенсинтазу, соответственно, ген PDR3 S. cerevisiae, кодирующий фактор транскрипции, мутантный ген ОВКТМ29 Haematococcus pluvialis, кодирующий β-каротин кетолазу сам по себе и с якорной группой к плазматической мембране. Путь синтеза регулировался температурно-чувствительной системой GAL (GAL4M9), чтобы отделить синтез продукта от роста клеток. Результирующий штамм продуцировал 19,2 мг/г сухого веса и 1,44 г/л кантаксантина после 250 часов двухэтапного культивирования в 5 литровом биореакторе на сложносоставных средах, содержащих пептон, дрожжевой экстракт, кукурузный экстракт, глюкозу и этанол и др.
Дрожжи Y. lipolytica являются привлекательным организмом-хозяином для производства каротиноидов [Abdel-Mawgoud А.М., Markham К.A., et al. (2018). Metab Eng, 50, 192-208. doi:10.1016/j.ymben.2018.07.016], имеют разработанный генетический инструментарий, а также имеют статус непатогенного и безопасного микроорганизма [Miller К.К., Alper Н.S. (2019). Appl Microbiol Biotechnol, 103(23-24), 9251-9262. doi: 10.1007/s00253-019-10200-х].
Группой авторов во главе с Zhiyong Cui [Cui Z., Zheng H., et al. (2021). Appl Environ Microbiol, 87(6). doi:10.1128/AEM.02666-20] получен штамм-продуцен кантаксантина Y. lipolytica несущий гены GGS1 Y. lipolytica, кодирующий геранилгеранилпирофосфат синтазу, CarRP и CarB Rhizomucor circinelloides, кодирующие бифункциональный фермент фитоен синтазу/ликопин β-циклазу (carRP) и фитоен дегидрогеназу (сагВ) и CrtW Brevundimonas sp., кодирующий β-каротин/зеаксантин-4-кетолазу. Результирующий рекомбинантный штамм при культивировании в малых объемах на среде с глюкозой в качестве источника углерода накапливал 0,036 г/л кантаксантина.
Описанное выше культивирование штамма дрожжей S. cerevisiae проводилось на сложносоставных средах при использовании в качестве источника углерода глюкозы, этанола и кукурузного экстракта. В литературе показано [Zhang S., Wang J., et al. (2021). Food Chem, 346, 128860. doi:10.1016/j.foodchem.2020.128860], что для биопроизводства этанола, пуллулана, фруктоолигосахаридов и т.д. в качестве дешевого источника углерода может быть использована меласса - побочный продукт сахарной промышленности, на 40-60% об./об. состоящий из сахарозы. Известно, что природные изоляты дрожжей Y. lipolytica не способны утилизировать сахарозу в качестве единственного источника углерода. Однако, в литературе описаны подходы к повышению эффективности утилизации сахарозы с помошью экспрессии гена ScSUC2 S. cerevisiae, кодирующего инвертазу, в сочетании с геном НХК1, кодирующем гексокиназу [Taratynova М.О., Kosikhina I.М., et al. (2021). Biotechnology, 37(3), 29-41. doi:10.21519/0234-2758-2021-37-3-29-41], а также транспортеров гексозы [Hapeta P., Szczepanska P., et al. (2021). Int J Mol Sci, 22(17). doi:10.3390/ijms221792828].
Расширение сырьевой базы для производства каротиноидов и конструирование рекомбинантных микроорганизмов с повышенным уровнем продукции каротиноида кантаксантина является актуальной задачей.
Технической проблемой, на решение которой направлено представленное изобретение является расширение арсенала микроорганизмов, продуцирующих каротиноид кантаксантин.
Раскрытие сущности итзобретения
Техническим результатом заявляемого изобретения является получение трансформанта дрожжей Yarrowia lipolytica, продуцирующий каротиноид кантаксантин.
Техническим результатом заявляемого изобретения является получение рекомбинантного штамма дрожжей Y. lipolytica - продуцента кантаксантина.
Технические результаты достигаются тем, что предложен трансформант дрожжей Yarrowia lipolytica, продуцирующий каротиноид кантаксантин содержащий в составе хромосомы, по крайней мере, по одной копии каждого из следующих генов, ген CarB М. circinelloides, кодирующий фитоендегидрогеназу, ген CarRP М. circinelloides, кодирующий бифункциональный фермент фитоен синтазу/ликопин β-циклазу, ген HMGR1 Y. lipolytica, кодирующий фермент 3-гидрокси-3-метилглютарил-кофермент А редуктазу, ген ERG20 Y. lipolytica, кодирующий фарнезилпирофосфатсинтазу, ген ERG12 Y. lipolytica, кодирующий мевалонаткиназу, нуклеотидную последовательность CarRP-GGPPs7, кодирующую полипептид, полученный в результате объединения бифункционального фермента фитоен синтазы/ликопин β-циклазы carRP М. circinelloides и геранилгеранилпирофосфат синтазы GGPPs7 Synechococcus sp, нуклеотидную последовательность CarRP-ERG20(F88C), кодирующую полипептид, полученный в результате объединения carRP М. circinelloides и модифицированного варианта фарнезилпирофосфатсинтазы ERG20F88C Y. lipolytica. кодон-оптимизированный ген НрВКТ Haematococcus pluvialis, кодирующий β-каротин кетолазу, нуклеотидная последовательность которого приведена в перечне последовательностей под номером SEQ ID NO:1, кодон-оптимизированный ген SpHXKl Schizosaccharomyces pombe, кодирующий гексокиназу, нуклеотидная последовательность которого приведена в перечне последовательностей под номером SEQ ID NO:2, кодон-оптимизированный ген YHT1 Y. lipolytica, кодирующий транспортер гексозы, нуклеотидная последовательность которого приведена в перечне последовательностей под номером SEQ ID NO:3, кодон-оптимизированный ген YHT3 Y. lipolytica, кодирующий транспортер гексозы, нуклеотидная последовательность которого приведена в перечне последовательностей под номером SEQ ID NO:5, кодон-оптимизированный ген YHT4 Y. lipolytica, кодирующий транспортер гексозы, нуклеотидная последовательность которого приведена в перечне последовательностей под номером SEQ ID NO:7.
Предложен рекомбинантный штамм дрожжей Y. lipolytica ВКПМ Y-5144 - продуцент кантаксантина.
Краткое описание чертежей
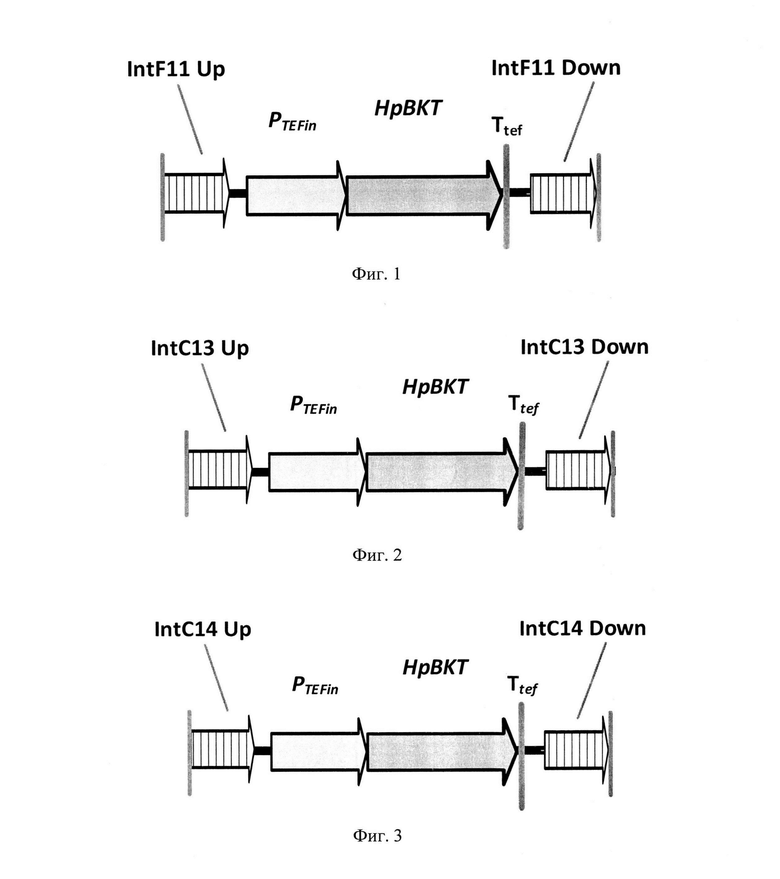
На фиг. 1 показана схема экспрессионной кассеты IntF11-PTЕFin-HpBKT.
На фиг. 2 показана схема экспрессионной кассеты IntC13-PTЕFin-HpBKT.
На фиг. 3 показана схема экспрессионной кассеты IntC14-PTЕFin-HpBKT.
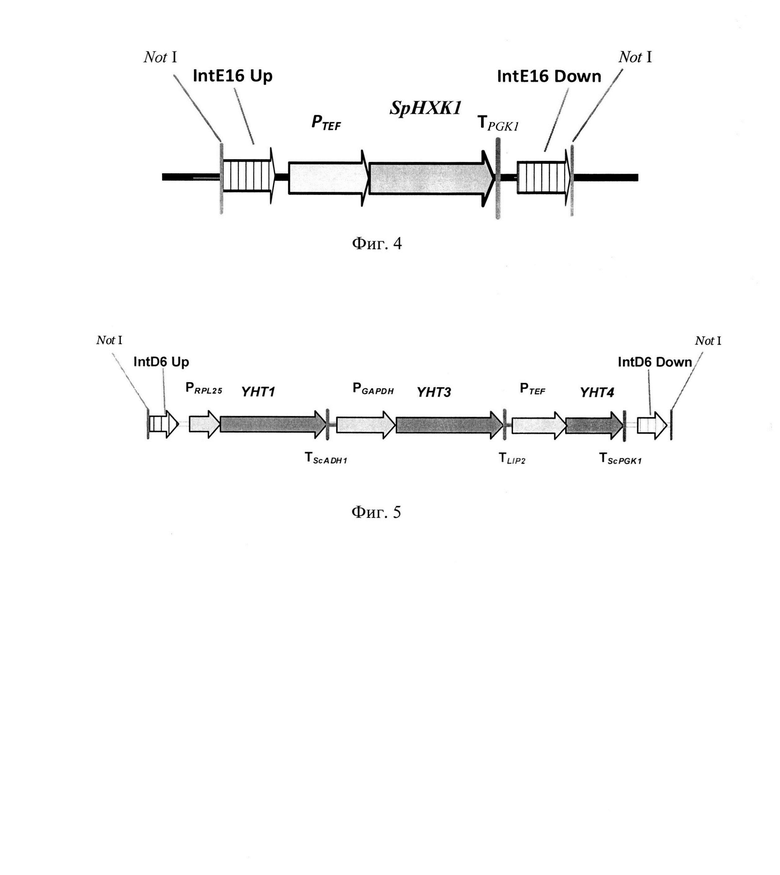
На фиг. 4 показана схема экспрессионной кассеты IntE16-SpHXKl.
На фиг. 5 показана схема экспрессионной кассеты IntD6-YHTl-YHT3-YHT4.
Осуществление изобретении
Конструирование экспрессионных кассет при получении трансформанта дрожжей осуществляют стандартными методами генетической инженерии [Sambrook J., Maniatis Т., Fritsch Е. Molecular Cloning: Laboratory Mannual, 2nd edition. New York: Cold Spring Harbor Laboratory Press, 1989] с использованием генетических элементов, подходящих для работы с дрожжами рода Yarrowia. В качестве промоторов могут быть использованы АСС1, ACL2, DGA1, GAPDH, GAPDHin, GPAT, GPD1, GPM1, ЕХР1, EYD1, EYK1, FBA1, FBAlin, FASa, FASBBBBB, HP4D, ICL1, IDH2, IDP2, ILV5, LEU2, LEUm, POT1, POX2, POX4, RPS7, TDH1, TEF1, TEFin, XPR2, YAT1, ZWF [Park Y. K, Korpys P., et al. (2019). FEMS Yeast Res, 19(1). doi:10.1093/femsyr/foy105, Kamineni A., Chen S., et al. (2020). FEMS Yeast Res, 20(5). doi:10.1093/femsyr/foaa035, 12. US20060115881, Madzak C, Treton В., et al. (2000). J Mol Microbiol Biotechnol, 2(2), 207-216, Tai M., Stephanopoulos G. (2013). Metab Eng, 15, 1-9. doi:10.1016/j.ymben.2012.08.007, Darvishi F, Ariana M., et al. (2018). Appl Microbiol Biotechnol, 102(14), 5925-5938. doi:10.1007/s00253-018-9099-x].
В качестве терминаторов транскрипции могут быть использованы Асо3, CYC1, Guo, LIP1, LIP2, РЕХЗ, РЕХ16, РЕХ20, РН05, TEF1, Tsynth2, Tsynth7, Tsynth8, Tsynth10, Tsynth22, Tsynth27, Tsynth30, XPR2 [Darvishi F., Ariana M., et al. (2018). Appl Microbiol Biotechnol, 102(14), 5925-5938. doi:10.1007/s00253-018-9099-x, Curran K.A., Morse N.J., et al. (2015). ACS Synth Biol, 4(7), 824-832. doi:10.1021/sb5003357, Larroude M., Park Y.K., et al. (2019). Microb Biotechnol, 12(6), 1249-1259. doi:10.1111/1751-7915.13427 и ЕР 2649 887 В1].
Введение генов может быть осуществлено как хромосомальной, так и эписомальной интеграцией, в качестве сайтов для гомологичной интеграции могут быть использованы последовательности, гомологичные участкам хромосомы дрожжей рода Yarrowia. В целях повышения эффективности экспрессии мРНК с интегрированных последовательностей при трансляции предпочтительно использовать гетерологичные гены, кодон-оптимизированные для дрожжей Y. lipolytica.
Трансформацию экспрессионной кассеты в клетки дрожжей Y. lipolytica осуществляют любым подходящим способом, в частности, методом электоропорации [Yeast Metabolic Engineering: Methods and Protocols, Methods in Molecular Biology. 2014, 1152, 43-62. doi:10.1007/978-1-4939-0563-8_3] или методом с использованием ацетата лития [Berardi Е., Thomas D.Y. (1990) Current Genetics 18, 169-170. doi:10.1007/BF00312606].
Пример 1. Получение кодон-оптимизированных генов и промоторов
Здесь и далее все гены кодон-оптимизируют для Y. lipolytica с помощью онлайн программы GenSmart™ [https://www.genscript.com/gensmart-free-gene-codon-optimization.html]. Все нуклеотидные последовательности, представленные ниже, синтезируют в виде фрагментов ДНК (синтез осуществлен компанией «Twist Bioscience Corporation (США)).
SEQ ID NO:1 - кодон-оптимизированный ген HpBKTHaematococcus pluvialis, кодирующий β-каротин кетолазу.
SEQ ID NO:2 - кодон-оптимизированный ген SpHXKl
Schizosaccharomyces pombe, кодирующий гексакиназу.
SEQ ID NO:3 - кодон-оптимизированный ген YHT1 Y. lipolytica, кодирующий транспортер гексозы.
SEQ ID NO:4 - промотор RPL25 белковой субъединицы 60S рибосомы L25 Y. lipolytica.
SEQ ID NO:5 - кодон-оптимизированный ген YHT3 Y. lipolytica, кодирующий транспортер гексозы.
SEQ ID NO:6 - промотор гена GAPDH Y. lipolytica, кодирующего глицеральдегид-3-фосфатдегидрогеназу.
SEQ ID NO:7 - кодон-оптимизированный ген YHT4 Y. lipolytica, кодирующий транспортер гексозы.
Пример 2. Получение трансформанта, продуцирующего кантаксантин
Получение трансформанта осуществляют путем поэтапного направленного введения генетических модификаций в штамм-реципиент, в качестве которого используют штамм линии W29 Y. lipolytica ВКПМ Y-4950 который имеет МАТа Δpex10 Aku70::Cas9 ZWF1 АСВР АСС1 DGA1 ScSUC2 НХК1 IntE::CarRP CarB GGS1 IntF14::HMG1 IntD4::.ERG12 lntA2::ERG20 lntC4::fusCarRP-GGPPs7 lntBl::CarB lntA4::fusCarRP-ERG20F88C.
Способ получения штамма Y. lipolytica ВКПМ Y-4950 и нуклеотидные последовательности CarRP-GGPPs7 и CarRP-ERG20(F88C) приведены в [RU2794980], нуклеотидные последовательности генов СагВ и CarRP М. circinelloides кодон-оптимизированных для Y. lipolytica приведены в [http://www.biotech-jr.ru/suppl/Suppl_37_3_Taratynova_ru.doc].
2.1 Интеграция в геном штамма-реципиента гена НрВКТ Haematococcus pluvialis, кодирующего β-каротин кетолазу.
Как описано в [RU2794980] конструируют экпрессионную кассету IntF11-PTЕFin-HpBKT (фиг. 1), которая имеет:
- участки гомологии IntF11 Up и IntF11 Down для интеграции в локус IntF11 на хромосоме F (IntF11 Up гомология к положению 3458258-3458790 пн, IntFl 1 Down к 3459091-3459592 пн);
- промотор гена TEF, кодирующего фактор элонгации трансляции Y. lipolytica, содержащий интрон (TEFin);
- кодон-оптимизированный ген НрВКТ Haematococcus pluvialis, кодирующий β-каротин кетолазу;
- терминатор гена TEF.
Интеграцию экспрессионной кассеты с геном НрВКТ Н. pluvialis (последовательность которого приведена в перечне последовательностей под номером SEQ ID NO:l) производят в локус IntF11 на хромосоме F (положение 3458258 - 3459592 пн) с помощью CRISPR-Cas9 системы [Yuzbashev Т., Yuzbasheva Е., et al. (2022). doi:10.21203/rs.3.rs-1570357/vl]. Для этого ДНК кассету IntFl l-PTЕFin-HpBKT (фиг. 1) используют совместно со вспомогательной кольцевой плазмидой pCNR-sgIntF11 [RU2794980] для котрансформации штамма ВКПМ Y-4950. Трансформанты отбирают на среде YPSuc Nat (мас. %: дрожжевой экстракт - 1, пептон - 2, сахароза - 1, агар - 1, вода - остальное) с добавлением нурсеотрицина (0,025 мас. %) по устойчивости к антибиотику нурсеотрицину. У трансформантов изолируют геномную ДНК и проводят ПЦР-анализ на правильность интеграции генетической кассеты по праймерам:
ACCTGACTGTTTACATCCGC (IntF11-chrom-F)
TCAAGTTCTTGCACTCTGTTG (IntF11-chrom-R)
Получают трансформанты с кассетой, интегрированной в локус IntF11 на хромосоме F.
Полученные трансформанты культивируют в жидкой среде YPSuc2 (мас. %: дрожжевой экстракт - 1, пептон - 2, сахароза - 2, вода - остальное) с добавлением сахарозы 2 мас. % на третьи и 1 мас. % на четвертые сутки культивирования в пробирках (50 мл) с рабочим объемом 10 мл при 30°С и постоянном перемешивании (250 об/мин) в течение 5 суток. Для определения сухого веса биомассы 1 мл культуральной жидкости центрифугируют при 18 000 g, осадок промывают и лиофилизируют в течение 24 ч с помощью системы FreeZone 6 Plus (Labconco, США).
Проводят экстракцию из лиофилизованных клеток. Для этого к 10-15 мг лиофилизованного образца добавляют 50 мкл стеклянных шариков диаметром 425-600 мкм (Sigma-Aldrich) и 500 мкл ацетона («ясхим», Россия), содержащего 0,01% 2,6-дитретбутил-4-метилфенола. Образцы гомогенизируют с использованием гомогенизатора Precellys 24 (Bertin, Франция) три раза при 5500 об/мин по 20 с × 2 цикла, центрифугируют при 18000 g и собирают ацетоновый экстракт. Процедуру экстракции клеток проводят трижды. Полученный объединенный экстракт анализируют методом ВЭЖХ на системе Waters (Waters, США), оснащенной колонкой WAT086344 С18 (3,9×150 мм, 4 мкм, 60А, 1/рк) (Nova-Рак, Украина). Условия ВЭЖХ: скорость потока - 1,0 мл/мин; мобильная фаза - ацетон: вода: метанол в соотношении 90:10:10 об./об. («ясхим»); детекция при 470 нм. β-каротин в исследуемом образце идентифицировали и определяли его количество соответственно по времени удерживания и калибровочному графику, построенному для стандартного образца кантаксантина (Sigma-Aldrich, США).
После экстракции из лиофилизированной биомассы по результатам ВЭЖХ отбирают трансформант 2.1.1 с лучшей продукцией кантаксантина (таблица 1).

2.2 Интеграция гена НрВКТН. pluvialis.
Интеграцию гена осуществляют в трансформант 2.1.1, полученный в примере 2.1.
Как описано в [RU 2794980] конструируют экпрессионную кассету IntC13-PTЕFin-HpBKT (фиг. 2), которая имеет:
- участки гомологии IntC13 Up и IntC13 Down для интеграции в локус IntC13 на хромосоме С (IntC13 Up гомология к положению 2646315-2646874 пн, IntC13 Down к 2647563-2648077 пн);
- промотор гена TEF, кодирующего фактор элонгации трансляции Y. lipolytica, содержащий интрон (TEFin);
- кодон-оптимизированный ген НрВКТ Haematococcus pluvialis, кодирующий Р-каротин кетолазу;
- терминатор гена TEF.
Интеграцию экспрессионной кассеты с геном НрВКТ Н. pluvialis (последовательность которого приведена в перечне последовательностей под номером SEQ ID NO:1) производят в локус IntC13 на хромосоме С (положение 2646315 - 2648077 пн) с помощью CRISPR-Cas9 системы. Для этого ДНК кассету IntC13-PTЕFin-HpBKT (фиг.2) используют совместно со вспомогательной кольцевой плазмидой pCNR-sgIntC13 [RU2794980] для котрансформации трансформанта 2.1.1. Трансформанты отбирают на среде YPSuc Nat по устойчивости к антибиотику нурсеотрицину. У трансформантов изолируют геномную ДНК и проводят ПНР-анализ на правильность интеграции генетической кассеты по праймерам:
CCATGTGTCTATAAGTACAGC (IntC13-chrom-F)
TAACCCTGACAGTTTGATAGC (IntC13-chrom-R)
Получают трансформанты с кассетой, интегрированной в локус IntC13 на хромосоме С.
Полученные трансформанты культивируют как описано в примере 2.1. По примеру 2.1 отбирают трансформант 2.2.2 с лучшей продукцией кантаксантина (таблица 2).

2.3 Интеграция гена НрВКТН. pluvialis.
Интеграцию гена осуществляют в трансформант 2.2.2, полученный в примере 2.2.
Как описано в [RU 2794980] конструируют экпрессионную кассету IntC14-PTEFin-HpBKT (фиг. 3), которая имеет:
- участки гомологии IntC14 Up и IntC14 Down для интеграции в локус IntC14 на хромосоме С (IntC14 Up гомология к положению 3133312-3133958 пн, IntC14 Down к 3134647-3135244 пн);
- промотор гена TEF, кодирующего фактор элонгации трансляции Y. lipolytica, содержащий интрон (TEFin);
- кодон-оптимизированный ген НрВКТ Haematococcus pluvialis, кодирующий β-каротин кетолазу;
- терминатор гена TEF.
Интеграцию экспрессионной кассеты с геном НрВКТ Н pluvialis (последовательность которого приведена в перечне последовательностей под номером SEQ ID NO:1) производят в локус IntC14 на хромосоме С (положение 3133312 - 3135244 пн) с помощью CRISPR-Cas9 системы. Для этого ДНК кассету IntC14-PTЕFin-HpBKT (фиг.3) используют совместно со вспомогательной кольцевой плазмидой pCNR-sgIntC14 [RU 2794980] для котрансформации трансформанта 2.2.2. Трансформанты отбирают на среде YPSuc Nat по устойчивости к антибиотику нурсеотрицину. У трансформантов изолируют геномную ДНК и проводят ПЦР-анализ на правильность интеграции генетической кассеты по праймерам:
AACTTTCTGAAACTTGCTTTCC (IntC14-chrom-F)
AACCAAACACGTGATCAAAGG (IntC14-chrom-R)
Получают трансформанты с кассетой, интегрированной в локус IntC14 на хромосоме С.
Полученные трансформанты культивируют как описано в примере 2.1. По примеру 2.1 отбирают трансформант 2.3.2 с лучшей продукцией кантаксантина (таблица 3).

2.4 Интеграция гена SpHXKl Schizosaccharomyces pombe, кодирующего гексокиназу.
Интеграцию гена осуществляют в трансформант 2.3.2, полученный в примере 2.3.
Для этого конструируют экспрессионную плазмиду pE16US1.2-TEFp-SpHXK21-SdPGKA, содержащую ген SpHXKl S. pombe для интеграции в локус IntE16 на хромосоме Е (положение 3975493 - 3976833 пн) с помощью CRISPR-Cas9 системы.
Плазмиду pE16US1.2-TEFp-SpHXK1-ScPGK1 получают с использованием эндонуклеазы рестрикции Есо31I методом Golden Gate [Yuzbashev Т., Yuzbasheva Е., et al. (2022). doi:10.21203/rs.3.rs-1570357/vl] из следующих плазмид pYTK001-SpHXK1, pE16US1.2 (№175685, Addgene), pProC-TEFl (№175721, Addgene), pTerC-ScPGK1 (№175725, Addgene).
Для этого получают плазмиду pYTK001-SpHXKl как описано ниже.
На основе последовательности SEQ ID NO:2 получают фрагмент ScPGK1-GGA с помощью амплифицикации по паре праймеров:
ATATCGTCTCATCGGGGTCT (GGA-F)
TATACGTCTCTGGTCGGTCT (GGA-R)
Фрагмент ScPGK1-GGA собирают с плазмидой pYTKOOl (№175709, Addgene) в единую плазмиду pYTK001-ScPGKl с использованием эндонуклеазы рестрикции Esp3l по методу Golden Gate.
ДНК кассету IntE16-SpHXKl (фиг. 4) получают путем обработки плазмиды pE16US1.2-TEFp-SpHXK1-ScPGKl эндонуклеазой рестрикции NotI и используют совместно со вспомогательной кольцевой плазмидой pCasNA-IntE16 (№175637, Addgene) для котрансформации трансформанта 2.3.2. Трансформанты отбирают на среде YPSuc Nat по устойчивости к антибиотику нурсеотрицину. У трансформантов изолируют геномную ДНК и проводят ПНР-анализ на правильность интеграции генетической кассеты по праймерам:
GCCTTGCAGAGCACAACTGGCAG (IntE16-chrom-F)
ACTCGGGAACTACGATAACTGCAAGTAGCG (IntE 16-chrom-R)
Получают трансформанты с кассетой, интегрированной в локус IntE16 на хромосоме Е.
Полученные трансформанты культивируют как описано в примере 2.1. По примеру 2.1 отбирают трансформант 2.4.2 с лучшей продукцией кантаксантина (таблица 4).

2.5 Интеграция генов YHT1, YHT3 и YHT4 Y. lipolytica, кодирующих транспортеры гексозы.
Интеграцию кассеты генов YHT1, YHT3 и YHT4 Y. lipolytica осуществляют в трансформант 2.4.2, полученный в примере 2.4, в локус IntD6 на хромосоме D (положение 1494593 - 1496287 пн) с помощью CRISPR-Cas9 системы.
Для этого получают плазмиду pD6UA2.3-PrPL25-YHT1-Tscadh1-Pgapdh-YHT3-TLIP2-YHT4-TScPGK1, которую собирают методом Golden Gate из следующих плазмид:
1. Плазмида IntD6-PRPL25-YHT1-TsCADH1.
На основе последовательности SEQ ID NO:3 получают фрагмент YHT1-GGA с помощью амплифицикации по паре праймеров GGA-F / GGA-R. Фрагмент YHT1-GGA собирают с плазмидой pYTK001 в единую плазмиду pYTK001-YHT1 с использованием эндонуклеазы рестрикции Esp3I по методу Golden Gate.
На основе последовательности SEQ ID NO:4 получают фрагмент Prpl25-GGA с помощью амплифицикации по паре праймеров GGA-F / GGA-R. Фрагмент Prpl25-GGA собирают с плазмидой pYTK001 в единую плазмиду PYTK001-Prpl25 с использованием эндонуклеазы рестрикции Esp3I по методу Golden Gate.
Из полученных плазмид: pD6US1.l (№175678, Addgene), pYTK001-Prpl25, pYTK001-YHT1, pTerC-ScADH1 (№175724, Addgene) с использованием эндонуклеазы рестрикции Есо31I конструируют единую экспрессионную плазмиду IntD6-PRPL25-НРЕ1-TScADH1.
2. Плазмида lntD6-PGAPDH-YHT3-TLIP2.
На основе последовательности SEQ ID NO:5 получают фрагмент YHT3-GGA с помощью амплифицикации по паре праймеров GGA-F / GGA-R. Фрагмент YHT3-GGA собирают с плазмидой pYTK001 в единую плазмиду pYTK001-YHT3 с использованием эндонуклеазы рестрикции Esp31 по методу Golden Gate.
На основе последовательности SEQ ID NO:6 получают фрагмент GAPDH-GGA с помощью амплифицикации по паре праймеров GGA-F / GGA-R. Фрагмент GAPDH-GGA собирают с плазмидой pYTK001 в единую плазмиду pYTK001-GAPDH с использованием эндонуклеазы рестрикции Esp31 по методу Golden Gate.
Из полученных плазмид: pD6US1.2 (№175679, Addgene), pYTK001-GAPDH, pYTK001-YHT3, pTerC-LIP2 (№175723, Addgene) с использованием эндонуклеазы рестрикции Есо31I конструируют единую экспрессионную
плазмиду IntD6-PGAPDH-YH3-TLIP2.
3. Плазмида IntD6-PTEF-YHT4-TScPGK1.
На основе последовательности SEQ ID NO:7 получают фрагмент YHT4-GGA с помощью амплифицикации по паре праймеров GGA-F / GGA-R. Фрагмент YHT4-GGA собирают с плазмидой pYTK001 в единую плазмиду pYTK001-YHT4 с использованием эндонуклеазы рестрикции Esp3I по методу Golden Gate.
Из полученных плазмид: pD6US1.3 (№175680, Addgene), pProC-TEFl (№175721, Addgene), pYTK001-YHT4, pTerC-ScPGK1 (№175725, Addgene) с использованием эндонуклеазы рестрикции Есо31I конструируют единую экспрессионную плазмиду IntD6-PTEF-YHT4-TScpGK1.
Единую экспрессионную плазмиду pD6UA2.3-PRPL25-YHT1-Tscadh1-Pgapdh-YHT3-TLIP2-PTef-YHT4-TscPg1 собирают из четырех плазмид pD6UA2.3 (№175616, Addgene), IntD6-PRPL25-YHY1-TScADH1, IntD6-РGарDн-YHT3-ТLIP2, IntD6-PTEF-YHT4-TScPGK1 c использованием эндонуклеазы рестрикции Esp3I.
Вспомогательную плазмиду pCasNA-IntD6 для осуществления интеграции генов YHT1, YHT3 и YHT4 с помощью CRISPR-Cas9 системы конструируют с помощью олигонуклеотида
TCGATGGGCCCCCGGTTCGATTCCGGGTCGGCGCAAAGCTTAATAAACAGCAAAGGTTTTAGAGCTAGAAATAGCAAGTTAAAATAAGGC (IntD6b-20bp-N).
Фрагмент ДНК IntD6-YHT1-YHT3-YHT4 (фиг. 5) получают путем обработки плазмиды pD6UA2.3-PrpL25-YHT1-TScadh1-Pgapdh-YHT3-ТLIр2-Ртер-YHT4-TScPGK1 эндонуклеазой рестрикции Noil и используют совместно со вспомогательной кольцевой плазмидой pCasNA-IntD6 для ко-трансформации трансформанта 2.4.2. Трансформанты отбирают на среде YPSuc Nat по устойчивости к антибиотику нурсеотрицину. У трансформантов изолируют геномную ДНК и проводят ПЦР-анализ на правильность интеграции генетической кассеты по праймерам: AGGTGGTCAATGAGTTGTCG (IntD6-chrom-F) TACGTGTGTTTGGCTGTCTC (IntD6-chrom-R)
Получают трансформанты с кассетой, интегрированной в локус IntD6 на хромосоме D. Полученный трансформант имеет генотип МАТа Apex 10 Δkux10::Cas9 ZWF1 АСВР АСС1 DGA1 ScSUC2 HXK1 IntE1::CarRP CarB GGS1 IntF14::HMG1 lntD4::ERG12 IntA2::ERG20 IntC4::fusCarRP-GGPPs7 InXBl::CarB IntA4::fusCarRP-ERG20F88C IntF11::HpBKT UntC13::НрВКТ IntC14::НрВКТ UntE16::SpHXKl IntD6::YHT1 YHT3 YHT4 и продуцирует кантаксантин на среде с мелассой в качестве единственного источника углерода.
Полученные трансформанты культивируют как описано в примере 2.1. По примеру 2.1 отбирают трансформант 2.5.2 с лучшей продукцией кантаксантина (таблица 5).

Отобранный трансформант депонирован в Биоресурсном Центре Всероссийская Коллекция Промышленных Микроорганизмов (БРЦ ВКПМ) НИЦ «Курчатовский институт» (117545 Москва, 1-ый Дорожный пр-д, д. 1) как штамм дрожжей Y. lipolytica ВКПМ Y-5144.
Признаки штамма дрожжей Y. lipolytica ВКПМ Y-5144 Культурально-морфологические признаки.
Суточная культура в жидкой YPD среде (мас. %: дрожжевой экстракт - 1, пептон - 2, глюкоза - 1, вода - остальное) представлена овальными, удлиненно-овальными, округлыми клетками размером 4,6-6,0×4,5-12,5 мкм. Почкование полярное или латеральное, на узком основании. К третьим суткам большинство клеток почкуются и образуют истинный и псевдомицелий.
При росте в жидкой среде YPD при 28°С в течение 24 ч культивирования - жидкость мутная, осадок красный, коагуляции не наблюдается, пристеночных пленок не образует.
Клетки хорошо растут на простых питательных средах. Колонии на мальтоагаре (возраст 1 неделя) красного цвета, пастообразные, слегка приподнятые в центре, морщинистые, с фестончатым краем. Штрих на мальтоагаре непрерывный, плоский, блестящий, красного цвета, пастообразный, края ровные, со временем становится складчатым. Спор не образует.
Физиолого-биохимические признаки.
Облигатный аэроб. Сахара не сбраживает. Ассимилирует: сахарозу, глюкозу, D-галактозу (медленно), L-сорбозу, D-рибозу, этанол, глицерин, эритрит, адонит, D-маннит, сорбит и молочную, янтарную, лимонную, глюконовую кислоты. Не ассимилирует: мальтозу, лактозу, целлобиозу, трегалозу, мелибиозу, раффинозу, мелицитозу, инулин, крахмал, D-ксилозу, L- и D-арабинозу, раммозу, дульцит, инозит, D-глюкозамин и глюкуроновую, 2-кетоглюконовую, 5-кетоглюконовую кислоты. Не ассимилирует нитраты, жирные кислоты и алканы. Не растет в безвитаминной среде, требует присутствия в среде тиамина, не требует биотина.
Оптимальное значение рН для роста 5,5-7,0. Не растет при 37°С. Максимальная температура роста 35°С. Разжижает желатин. Гидролизует мочевину.
Пример 3. Культивирование штамма дрожжей Y. lipolytica ВКПМ Y-5144 в ферментере на среде, содержащей мелассу в качестве единственного источника углерода.
Культивирование штамма дрожжей Y. lipolytica ВКПМ Y-5144 проводят в 3-л ферментере модель КФ-103 производства ООО-фирма "Проинтех" (Пущино, РФ) с рабочим объемом 1 л при температуре 29°С в среде следующего состава (мас. %: дрожжевой экстракт - 2, пептон - 4, меласса - 1). Концентрированный раствор, содержащий меласса (мас. % - 5), готовят отдельно и вносят со скоростью 8,4 г/(л ч) начиная с 16 часа культивирования.
В качестве пеногасителя используют адеканоль (0.1% v/v). Раствор 25% водного аммиака используют для поддержания значения рН среды равного 5,5. Уровень растворенного в среде кислорода составляет не менее 25%. Подготовку посевной культуры проводят в три этапа:
- пересев штамма на агаризованную среду YPD (мас. %: дрожжевой экстракт - 1, пептон - 2, глюкоза - 1, агар - 1, вода - остальное)
- посев в жидкую среду YPD в пробирку с общим объемом 50 мл и рабочим объемом 5 мл и инкубация в течение 24 ч при 30°С и постоянном перемешивании (250 об/мин).
- посев в жидкую среду YPD в колбы с общим объемом 750 мл и рабочим объемом 50 мл и инкубация в течение 24 ч при 30°С и постоянном перемешивании (250 об/мин).
Посевная культура вносится в ферментер в количестве 10% от рабочего объема. Отбор проб осуществляется каждые 24 ч.
Сконструированный штамм дрожжей Y. lipolytica ВКПМ Y-5144 в ферментере накапливает биомассу 61,6 г сухого веса/л и продуцирует 14,4 мг/г сухого веса и 887,0 мг/л кантаксантина за 120 часов культивирования на среде, содержащей мелассу в качестве единственного источника углерода.
Данные культивирования показывают, что штамм дрожжей У. lipolytica ВКПМ Y-5144 эффективно растет при использовании мелассы, в качестве единственного источника углерода. На данном дешевом субстрате штамм продуцирует 0,9 г/л кантаксантина всего за 120 часов культивирования. Данный результат в 25 раз превосходит титр, описанный ранее [Cui Z., Zheng Н., et al. (2021). Appl Environ Microbiol, 87(6). doi:10.1128/AEM.02666-20] на дрожжах Y. lipolytica (0,036 г/л).
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing originalFreeTextLanguageCode="ru"
nonEnglishFreeTextLanguageCode="ru" dtdVersion="V1_3"
fileName="Transformant Yarrowia lipolytica is a producer of
canthaxanthin.xml" softwareName="WIPO Sequence"
softwareVersion="2.1.2" productionDate="2023-08-15">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText></ApplicationNumberText>
<FilingDate></FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>53266309</ApplicantFileReference>
<ApplicantName languageCode="ru">Национальный исследовательский
центр "Курчатовский институт"</ApplicantName>
<ApplicantNameLatin>National Research Center "Kurchatov
Institute"</ApplicantNameLatin>
<InventionTitle languageCode="ru">Трансформант дрожжей Yarrowia
lipolytica, продуцирующий кантаксантин</InventionTitle>
<SequenceTotalQuantity>7</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>1040</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..1040</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Yarrowia lipolytica</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cacgtcatccgaccagcactttttgcagtactaaccgcagcacgtggcc
tctgctctgatggtcgagcagaagggctctgaggccgctgcctcttctcccgacgtgctgcgagcctggg
ctacccagtaccacatgccttccgagtcctctgacgccgctcgacccgctctgaagcacgcctacaagcc
tccagcctccgacgccaagggcatcaccatggctctgaccatcatcggaacctggaccgccgtgttcctg
cacgccatcttccagattcgactgcccacctctatggaccagctgcactggctgcccgtgtctgaggcca
ccgctcagctgctcggcggatcttcttctctgctgcacattgccgccgtctttatcgtgctcgagttcct
gtacaccggcctgttcatcaccactcacgacgccatgcacggcaccattgctctgcgacaccgacagctg
aacgacctgctgggcaacatctgcatctccctgtacgcctggttcgactactctatgctgcgacgaaagc
actgggagcaccacaaccacaccggcgaggtcggcaaggaccccgacttccacaagggcaaccccggact
ggtgccctggttcgcctctttcatgtcctcttacatgtctctgtggcagttcgcccgactggcctggtgg
gccgtcgtcatgcagatgctgggcgctcccatggccaacctgctggtgttcatggccgctgctcccatcc
tgtccgccttccgactgttctacttcggcacctacctgcctcacaagcccgagcctggacctgccgccgg
atctcaggacatggcctggttccgagccaagacctctgaggcttctgacgtgatgtctttcctgacctgc
taccacttcgacctgcattgggagcatcatcgatggccctacgctccctggtggcagctccctcactgcc
gacgactgtctggccgaggactggtccccgctctggcctaagctgcttgtacctagtgcaaccccagttt
gttaaaaatta</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>1504</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..1504</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Yarrowia lipolytica</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atatcgtctcatcggggtctcaaatgtctctgcatgacgcctaccattg
gccctctcgaaccccttctagaaaaggctccaacatcaagctgaataagactctccaggaccacctggat
gagttggaggagcagtttactatcccaactgaactgctgcaccgagtgactgaccgtttcgtgtcggaat
tgtataagggtcttaccaccaaccccggcgacgtccccatggtccctacctggatcattggaactcccga
cggaaacgagcacggcagttatctggcgctggatttgggaggtacaaatctccgggtctgcgctgttgaa
gtccagggcaacggtaaatttgatatcacccaatccaagtatcgcttaccccaggagctgaaggttggca
cgcgtgaggcactgtttgactacattgccgactgtattaagaagtttgtggaggaggtgcatccaggtaa
atcgcagaacctcgagattggtttcaccttttcctacccttgtgtgcagcgatccatcaacgacgcttcg
ctggttgcctggacgaagggcttcgatattgatggggtcgagggcgaatctgtcggccctcttctcagcg
ctgccctaaaacgagtaggctgcaacaacgtcaggctcaacgccatactgtctgataccactggaacgtt
agttgcaagcaactacgcctctccgggtaccgagattggagtcatctttggaaccggttgcaatgcttgc
tatatagagaagttctctgagattcccaagctccacaagtacgacttcccggaggacatgaacatgatca
tcaattgcgagtggtgtgactttgacaaccagcacgtggtactaccccggaccaagtacgatgtggcgat
cgacgaggaaagtccaagacctggtctgcaaacttacgaaaagatgattgctggctgctacctgggagac
attctgcgacgaatcctgctcgacctgtacgagcaaggagcactcttcaacggccaggatgttactaaaa
ttcgagatcccctcgccatggacacatcggtcctttcggccattgaggttgaccccttcgagaacctgga
cgagactcagaccttgtttgaggagacttacggccttaaaacgacagaagaggagcgacagttcatccga
cgagcctgcgagctcattgggacaagaagcgcgagactctcagcttgtggtgtgtgtgccctcgtgcgaa
agatgaataagccttccatgatcgttgggaccgacgggagcgtgtacaacctgtaccctcggttcaagga
ccgactagctcaggccttcaaggatattcttggagaagagatcggctcaaaggtggtcaccatccctgca
gaggatggatccggtgttggagctgccttggtgtccgcactagaggccaagggaaaggctctcacatccg
acattcttgctgagcatctgaagaactaatctatgagaccgaccagagacgtata</INSDSeq_seque
nce>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>1597</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..1597</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q3">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Yarrowia lipolytica</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atatcgtctcatcggggtctcaaatggggttggccaacatcattaatag
aggtgagaagcccgagggctctgccttcatggctgcatttgttgcggtgtttgtggcctttggaggcatt
ctcttcggctatgacacaggaacgatctctggtgtcatggctatgcccttcgtcaaaaagacatttaccg
acgatggactagaatttacgagtgagcagacctctctgatcacatcaattttgagtgccgggactttcac
aggtgcgattagtgctccctgggcttccgatacgctgggccgaaggctcggactcatccttttctgcgtg
gtcttttctgtgggagccatcctgcagactgcggcgactggtagaactctactcatcgtgggacgagtcg
ttgcgggccttggcgttgggggtgtctcgtccattgtgccgctgtaccagtccgaggtggcaccaaagtg
gattcgaggagctgtagtgtccatctaccaattcgcaatcactattggacttctccttgctgccattgtc
aacaacgctaccaagaacaaggacaacagcgcttcgtaccgaatccctttgggcctgcaactgctctggg
ccgttattctgagcggaggcctgatcttgctgccggaaacccctcgattctggattaaaaagggcgagta
cgataaggctgccgactctttacgtcggctcagacggttacccgtggagcacgaagctgtgcagaaggaa
ctgctcgaaatacagtcgtcacacgaccacgagatgcagattggctcggccacatgggcggcatgtttct
ctcccaagggttcgcaactcaagcgaatgctgactggtattgccatccaggccctccagcagctgaccgg
catcaactttatcttctactacggcactgagttcttcaagaagtctaacatctccaaccctttcctgata
caaatgatcaccaacattgtgaacgtggtgatgaccatccctggaatcatgttcgtggaccgtgttggcc
gccgcaagctgttactcattggtgccatcgtcatgtgttcctcggagtttatcgtagctgctgtcggaac
cgccattgataacgagacttcttccaaggtgttgatagcattcacttgcaccttcattgctggcttcgca
gcaacctggggtccaattgcctgggtcgttattggagagattttccccctgcgaatccgagccaaaggtg
tcgctctgtgtgccgcctctaactggctgttcaactttgccatcgcgtttgccaccccctatcttgttga
tgaagcccctggaagcgccggcctcaagaccaaagtctttttcatttggggcggatgcaattttctgtgc
atcgctttcacctacttctttatttatgagactaagggtctaactctggaggaggtggaccagatgtacg
ccgagatcaagatcgcttcacgatctcaccagtttgttcctacaacgcgggtagcagcctacgacgagca
tgcttccgacgacaagaaggatggccagcatgtctacattgaatccgtttaatctatgagaccgaccaga
gacgtata</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>853</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..853</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Yarrowia lipolytica</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atatcgtctcatcggggtctcaaacggttcaaccgccactttcgcacct
ctggatgcaagaagccccacatcaccattgtcaacagagcaacggtagaagcccgaatgatggccgatag
atacgaatttcattagtaatgaacttgaatagtgtatctgcgtggcgggccttatacctgtgcttttatt
ccacaatgttcagtgagatattaacctgcagtacatccgcggttaaataagtttagttgggagtacttgt
tgtatgtcaacagacgacctactgactctgtattgtagggcaacagttcgcacaagtagacgtttagcac
attaatcttcgaagtaatgctttatcacgtgacataatctgcaaaaagtcatatgacgctcctgggcaat
aacatgcaacaagtccggagaataaggcccattatttagtgcatggcgatcttcgaacgcgcacttctat
agctccatccatccatcccatcaccaagtccaaactccgtctaccaataagggtttaaggttcgtccttt
ttggggtcatctatccacgtgacctattttagacgaccctaaatatagctctagaaatcgtcattatggt
tgaatgtttctgtccacatgccactacaagacaaatatttacctggtaataattcccctaccacatcttc
cgctcccacacagagttaccctgcctttagtctagggttagccctaaagacagtcacaggctaacctaaa
gcggtgccaaacttttcactacatcaaatccgccggctcaaaaatcttagttcctcagctcacactcacg
acatcaactctatgagaccgaccagagacgtata</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>1651</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..1651</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q5">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Yarrowia lipolytica</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atatcgtctcatcggggtctcaaatgtctacctctgccatgactgacga
ctaccccatggccgaggtttccatcaccgaagagactaagatgcctgagcctgccaagaaatcgcaactg
cttgttattcttctttgcctctttgcagcaatgggtggcttcgtgtttggttacgacaccggaactatca
gtggatttattaacatggacccttttttagaaagtttcggcgaggccgatgagtccggcgtatttttctt
gagccgaatcagggctggccttatcgtcggtctgttctccattggcgctttcctaggaacgctgctgggc
gggtttcttgctgacaaggtgggccggaagaagggtatcgtctacgtggccatggtctatattgttggca
tgctgatccagattacagctttcacagcctggtaccagatagcgatcgggcgagtgatcggtggtgtagg
aattggtgctctctctgtgctcgttcctatgttccagtccgaaacggcccctcagaacctgagaggtgct
ctggtgtcctccttccaactcttcatcactctcggaatctttatcggcttctctgtttgctacgccacca
agtctcgcttggacaccggagcctatcgtattcccatgggcctttgttttgcatgggccttcatattact
cattggtatgtgtttcatgcccgagtctcctcggtttctcgtgtctattggacgtatcgaggaggctcgg
aaagcaatggccatgaccaaccaagttccgctgcacgatgcagttattgacgaggagctcaaggcgattg
agaattctgtcatccgagagaagtcagcaggcaaggccacatggaaggagctgttcactggcgagccgag
aatgggatacagactgaccctaggcattttggtccaggtgctgcagcagctctgtggtgccaactacttt
ttctactacggaacatccattttcaaggcgattggaatgagcgattcgtttgccacttctatgatctttg
gaggaatcaacctgctgtctactttcggtggattgtacattgtggaccgattctgtcgacgaaagtgtct
gctaggaggagctatggtgatgtttgtctgcttcctggtgtactccactgtgggtttcgccgcactctat
cccaatggggacacgacactgccggcgcgcaaagctgtgggtgatgtcatgattctgtttacctgcattt
tcatagctgctttcgcctcgacctgggctcccattgcctttgtggttgtttcagaaaccttccccctgcg
aatgcgatccaagggcatggctgttgctacgggaggcaactggatgattaacttcctggtgtcgtttctc
actcccttcatcacctcatcgattggattcaagtacggctacgtgttcaccgcctgcatcggttttgcta
tcatcttcgtattcttctttatccccgaaacaaaaggcctgtcattggaggatgtcgatgaactctatgc
gtctggcgtgagcgcccgaaactcgccaaattgggtcccaagtagcaagaagcaggaagtcaacgagccc
aagaactccatgaccgacttttccgagtcgagctaatctatgagaccgaccagagacgtata</INSDSe
q_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>545</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..545</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Yarrowia lipolytica</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atatcgtctcatcggggtctcaaggactgcgtccgaaccagctccagca
gcgttttttccgggccattgagccgactgcgaccccgccaacgtgtcttggcccacgcactcatgtcatg
ttggtgttgggaggccactttttaagtagcacaaggcacctagctcgcagcaaggtgtccgaaccaaaga
agcggctgcagtggtgcaaacggggcggaaacggcgggaaaaagccacgggggcacgaattgaggcacgc
cctcgaatttgacacgagtcacggccccattcgcccgcgcaatggctcgccaacgcccggtcttttgcac
cacatcaggttaccccaagccaaacctttgtgttaaaaagcttaacatattataccgaacgtaggtttgg
gcgggcttgctccgtctgtccaaggcaacatttatataagggtctgcatcgccggctcaattgaatcttt
tttcttcttctcttctctatattcattcttgaattaaacacacatcaacatctatgagaccgaccagaga
cgtata</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>1714</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..1714</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q7">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Yarrowia lipolytica</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atatcgtctcatcggggtctcaaatgtacaaggtgcacaacccctatct
gactgctgctgtggccaccatgggaggcatgctttttggctttgatatcagctctgtttcggcttttgtc
ggcgaggataattacatgaactactttggtcatcccacctccttccaacagggtggcatcaccgctagta
tggccggtggaagcatgctgagctgtgcctttgccggctacatttccgaccgagtgggccgcaagcccac
tatccagttcgcggcagcttggtggatggttggagcttccattcaatgttcagctcagaacatgggccag
cttattgctgggagagccatttcagggctcggcataggactgggatcttctcagatccctgtgttcattt
ccgagctttcgcccaagaagattcgcggtcggctggtcggatgtttccagtggtctgtgacctggggaat
tctgataatgttctacatttcgtttggctgctcctacatcaagggccactcctctttccgtttggcctgg
ggtatccaactcatccctggtgccatgctggcgtttggcatgatgttgctggacgagtctcctcgatggc
tggcgtctaaggacagatgggaggaagccattcaaattattcgatcgatcaatgccaactatggcagcga
agaggacatcctcatggagattgaggacctacgggaggtggtgcgaatcgaccacgagtccaaaagtgta
acaatctgggatttgttccggaaagatagtatcaaccgtacgatggtgggtgtgtgggcccagatttggc
agcagctgaccggtatgaacatcatgatgtactacgtggttatcatcttcaagatggcgggttattcagg
caagtccgcagtcattgttagcggctccatccagtatattattaacgtggtcatgaccatccccgccctc
ttgttcatcgacaagatcggacgacgaccacttctgctgtgcggttctatgctcatggcaacttggcttc
tcgctgtcggaggaatgttaggagcctatggaattcagatgccccagggccttcctgctgtcccctccaa
aaaccaggccgccgacccctacaccaccatttacattcccgacaaccaagcaccagctcgaaaggcgatc
attgcctgctgctacctctttgttgcatcgtttgccccgacatggggtccggggatctggctctactgtt
ctgagattttccccaacaagcagcgagccctcgcaaattctctaactgctggtgccaactggggtttcaa
cttcgctctggctctctttgtgccaacggccttcaaaaatatcaactggaaggtctacatcatctttgga
gtgttctgcatcgtcatgtctattcatgtgtttttgctgttcccagaaacaaagggaaagtctctggagg
ttattgaccagatgtgggacgccagagttcctgcgtggaaaaccgcatcgtgggtccctgatcacatgcc
tagccactacgccggtgaccaggaggagaagccgacggatgagctggccgaggcgccgttccacgaagag
aacgctcccgtcaacacagagactcctccccatgaagatgagcctactttcgctgaaaccgagcccaaaa
cccagtaccctggaactgaacatgtttaatctatgagaccgaccagagacgtata</INSDSeq_seque
nce>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Модифицированная нуклеотидная последовательность, кодирующая полипептид, обладающий активностью фитоен синтазы и геранилгеранилпирофосфат синтазы (варианты), ее использование при конструировании трансформантов дрожжей Yarrowia lipolytica, продуцирующих каротиноиды | 2021 |
|
RU2794980C1 |
| Промотор для экспрессии генов и его использование при конструировании трансформанта дрожжей Yarrowia lipolytica, продуцирующего β-каротин | 2022 |
|
RU2819535C1 |
| Трансформант дрожжей Yarrowia lipolytica, продуцирующий линалоол | 2022 |
|
RU2809554C1 |
| Штамм дрожжей Yarrowia lipolytica - продуцент линалоола | 2023 |
|
RU2819537C1 |
| МУЛЬТИЗИМЫ И ИХ ИСПОЛЬЗОВАНИЕ В ПОЛУЧЕНИИ ПОЛИНЕНАСЫЩЕННЫХ ЖИРНЫХ КИСЛОТ | 2008 |
|
RU2517608C2 |
| ДЕЛЬТА-5-ДЕСАТУРАЗА И ЕЕ ПРИМЕНЕНИЕ ДЛЯ ПОЛУЧЕНИЯ ПОЛИНЕНАСЫЩЕННЫХ ЖИРНЫХ КИСЛОТ | 2007 |
|
RU2469092C2 |
| СПОСОБ ПОЛУЧЕНИЯ ЯНТАРНОЙ КИСЛОТЫ С ИСПОЛЬЗОВАНИЕМ ДРОЖЖЕЙ, ПРИНАДЛЕЖАЩИХ К РОДУ Yarrowia | 2009 |
|
RU2422526C2 |
| Трансформант Ogataea haglerorum - продуцент термостабильной α-амилазы | 2022 |
|
RU2795707C1 |
| ГЕНЕТИЧЕСКАЯ КОНСТРУКЦИЯ ДЛЯ ЭКСПОЗИЦИИ БЕЛКА НА ПОВЕРХНОСТИ КЛЕТОЧНОЙ СТЕНКИ ДРОЖЖЕЙ Yarrowia lipolytica | 2010 |
|
RU2451749C1 |
| Трансформант дрожжей Ogataea haglerorum, продуцирующий бета-маннаназу, содержащий в составе хромосомы синтетический ген MANS | 2020 |
|
RU2764793C1 |
Изобретение относится к области биотехнологии. Предложен трансформант дрожжей Yarrowia lipolytica, продуцирующий каротиноид кантаксантин и содержащий в составе хромосомы по крайней мере по одной копии каждого из следующих генов: ген CarB М. circinelloides, кодирующий фитоендегидрогеназу, ген CarRP М. circinelloides, кодирующий бифункциональный фермент фитоен синтазу/ликопин β-циклазу, ген HMGR1 Y. lipolytica, кодирующий фермент 3-гидрокси-3-метилглютарил-кофермент А редуктазу, ген ERG20 Y. lipolytica, кодирующий фарнезилпирофосфатсинтазу, ген ERG12 Y. lipolytica, кодирующий мевалонаткиназу, нуклеотидную последовательность CarRP-GGPPs7, кодирующую полипептид, полученный в результате объединения бифункционального фермента фитоен синтазы/ликопин β-циклазы carRP М. circinelloides и геранилгеранилпирофосфат синтазы GGPPs7 Synechococcus sp, нуклеотидную последовательность CarRP-ERG20(F88C), кодирующую полипептид, полученный в результате объединения carRP М. circinelloides и модифицированного варианта фарнезилпирофосфатсинтазы ERG20F88C Y. Lipolytica, кодон-оптимизированный ген НрВКТ Haematococcus pluvialis, кодирующий β-каротин кетолазу, нуклеотидная последовательность которого приведена в перечне последовательностей под номером SEQ ID NO: l, кодон-оптимизированный ген SpHXKl Schizosaccharomyces pombe, кодирующий гексокиназу, нуклеотидная последовательность которого приведена в перечне последовательностей под номером SEQ ID NO: 2, кодон-оптимизированный ген YHT1 Y. lipolytica, кодирующий транспортер гексозы, нуклеотидная последовательность которого приведена в перечне последовательностей под номером SEQ ID NO: 3, кодон-оптимизированный ген YHT3 Y. lipolytica, кодирующий транспортер гексозы, нуклеотидная последовательность которого приведена в перечне последовательностей под номером SEQ ID NO: 5, кодон-оптимизированный ген YHT4 Y. lipolytica, кодирующий транспортер гексозы, нуклеотидная последовательность которого приведена в перечне последовательностей под номером SEQ ID NO: 7. Также предложен рекомбинантный штамм дрожжей Y. lipolytica ВКПМ Y-5144 - продуцент кантаксантина. Изобретение обеспечивает расширение арсенала микроорганизмов, продуцирующих каротиноид кантаксантин. 2 н.п. ф-лы, 5 ил., 5 табл., 3 пр.
1. Трансформант дрожжей Yarrowia lipolytica, продуцирующий каротиноид кантаксантин, содержащий в составе хромосомы по крайней мере по одной копии каждого из следующих генов: ген CarB М. circinelloides, кодирующий фитоендегидрогеназу, ген CarRP М. circinelloides, кодирующий бифункциональный фермент фитоен синтазу/ликопин β-циклазу, ген HMGR1 Y. lipolytica, кодирующий фермент 3-гидрокси-3-метилглютарил-кофермент А редуктазу, ген ERG20 Y. lipolytica, кодирующий фарнезилпирофосфатсинтазу, ген ERG12 Y. lipolytica, кодирующий мевалонаткиназу, нуклеотидную последовательность CarRP-GGPPs7, нуклеотидную последовательность CarRP-ERG20(F88C), кодон-оптимизированный ген НрВКТ Haematococcus pluvialis, кодирующий β-каротин кетолазу, нуклеотидная последовательность которого приведена в перечне последовательностей под номером SEQ ID NO: 1, кодон-оптимизированный ген SpHXKl Schizosaccharomyces pombe, кодирующий гексокиназу, нуклеотидная последовательность которого приведена в перечне последовательностей под номером SEQ ID NO: 2, кодон-оптимизированный ген YHT1 Y. lipolytica, кодирующий транспортер гексозы, нуклеотидная последовательность которого приведена в перечне последовательностей под номером SEQ ID NO: 3, кодон-оптимизированный ген YHT3 Y. lipolytica, кодирующий транспортер гексозы, нуклеотидная последовательность которого приведена в перечне последовательностей под номером SEQ ID NO: 5, кодон-оптимизированный ген YHT4 Y. lipolytica, кодирующий транспортер гексозы, нуклеотидная последовательность которого приведена в перечне последовательностей под номером SEQ ID NO: 7.
2. Рекомбинантный штамм дрожжей Y. lipolytica ВКПМ Y-5144 - продуцент кантаксантина.
| Модифицированная нуклеотидная последовательность, кодирующая полипептид, обладающий активностью фитоен синтазы и геранилгеранилпирофосфат синтазы (варианты), ее использование при конструировании трансформантов дрожжей Yarrowia lipolytica, продуцирующих каротиноиды | 2021 |
|
RU2794980C1 |
| TRAMONTIN L.R.R | |||
| et al | |||
| Enhancement of Astaxanthin Biosynthesis in Oleaginous Yeast Yarrowia lipolytica via Microalgal Pathway | |||
| Microorganisms | |||
| Станок для придания концам круглых радиаторных трубок шестигранного сечения | 1924 |
|
SU2019A1 |
| Печь-кухня, могущая работать, как самостоятельно, так и в комбинации с разного рода нагревательными приборами | 1921 |
|
SU10A1 |
| LAZAR Z | |||
| et al | |||
| Characterization of hexose transporters in Yarrowia lipolytica reveals new groups of Sugar Porters | |||
Авторы
Даты
2024-09-30—Публикация
2023-11-01—Подача