Область техники
Настоящее изобретение относится к области метаболической инженерии и биотехнологии и может быть использовано для получения продуцента линалоола с помощью дрожжей Yarrowia lipolytica.
Уровень техники
Линалоол (2,6-диметил-2,7-октадиен-6-ол) представляет собой ароматический монотерпеновый спирт, который широко встречается в эфирных маслах растений в виде двух энантиомеров (S- и R-) и используется в парфюмерии, косметике, бытовых чистящих средствах и пищевых добавках [An, Q., et al., Recent updates on bioactiveproperties of linalool. Food Funct, 2021. 12(21): p.10370-10389].
Глобальный производственный рынок этого вещества оценивался в 9980 миллионов долларов США в 2019 году и, как ожидается, достигнет 12300 миллионов долларов США к 2024 году [Ferraz, С.А., et al., Isopentenol Utilization Pathway for the Production of Linalool in Escherichia coli Using an Improved Bacterial Linalool/Nerolidol Synthase. Chembiochem, 2021. 22(13): p.2325-2334]. В основном, линалоол получают при помощи химического синтеза [Caputi, L. and Е. Aprea, Use of terpenoids as natural flavouring compounds in food industry. Recent Pat Food Nutr Agric, 2011. 3(1): p. 9-16]. В качестве отдушки он широко применяется в производстве духов, лосьонов, мыла и шампуней, а также в некосметических бытовых продуктах, таких как моющие и чистящие средства. В свою очередь, в фармацевтической промышленности он используется как предшественник для синтеза витамина Е [Mązka, W., et al., Natural Compounds in the Battle against Microorganisms-Linalool. Molecules, 2022. 27(20)]. Линалоол обладает противовоспалительным и антиоксидантным, а также противораковым свойствами [Pereira, I., et al, Linalool bioactive properties and potential applicability in drug delivery systems. Colloids Surf В Biointerfaces, 2018. 171: p. 566-578, Elbe, H., et al., Anticancer activity of linalool: comparative investigation of ultrastructural changes and apoptosis in breast cancer cells. Ultrastruct Pathol, 2022. 46(4): p. 348-358 и Silva, B.I.M., et al., Anticancer activity of monoterpenes: a systematic review. Mol Biol Rep, 2021. 48(7): p. 5775-5785]. Также сообщается о его седативном, анксиолитическом и противосудорожном действии. Эффективность пищевых добавок, содержащих линалоол, при лечении тревожных расстройств подтверждена клиническими испытаниями [Kasper, S., et al., Efficacy and safety of silexan, a new, orally administered lavender oil preparation, in subthreshold anxiety disorder - evidence from clinical trials. Wien Med Wochenschr, 2010. 160(21-22): p. 547-56].
Линалоол содержится во многих растениях, из них в промышленности для получения этого вещества широко используются эфирные масла бразильского палисандра (Aniba rosaeodora) и лаванды {Lavendula angustifolia). Однако A. rosaeodora грозит полное исчезновение, а выход эфирного масла из L. angustifolia слишком низок [Cheng, В.-Н., et al., Potential Source of S-(+)-Linalool from Cinnamomum osmophloeum ct. linalool Leaf: Essential Oil Profile and Enantiomeric Purity. Journal of Agricultural and Food Chemistry, 2012. 60(31): p. 7623-7628].
Микробный синтез линалоола позволит избежать ограничений, связанных с использованием растительного сырья. В то же время, микробный линалоол, как и полученный из растений, может маркироваться как натуральный продукт, что выгодно отличает его от химически синтезированного, так как в последние годы натуральные продукты более предпочтительны на потребительском рынке [Schempp, F.M., et al., Microbial Cell Factories for the Production of Terpenoid Flavor and Fragrance Compounds. Journal of Agricultural and Food Chemistry, 2018. 66(10): p. 2247-2258]. Кроме того, микробный синтез дает возможность производить энантиоселективное производство линалоола, которое сложно осуществить с помощью химического синтеза, за счет использования стереоселективности (энантиоселективности) ферментативных реакций [Nitta, N., et al., Fermentative production of enantiopure (S)-linalool using a metabolically engineered Pantoea ananatis. Microb Cell Fact, 2021. 20(1): p. 54]. Стереоселективность имеет значение в промышленности, поскольку два существующих в природе энантиомера линалоола имеют разную силу и профиль аромата, а также обладают различными биологическими свойствами [Nitta, N., et al., Fermentative production of enantiopure (S)-linalool using a metabolically engineered Pantoea ananatis. Microb Cell Fact, 2021. 20(1): p.54]. Так, интенсивность аромата S-изомера примерно в 9 раз выше, чем R-изомера [Aprotosoaie, А.С, et al., Linalool: a review on a key odorant molecule with valuable biological properties. Flavour and Fragrance Journal, 2014. 29(4): p. 193-219].
На данный момент есть данные о получении линалоола с помощью микробиологического синтеза в таких организмах как Pantoea ananatis, Escherichia coli, Saccharomyces cerevisiae, Y. lipolytica [Taratynova, M.O., et al., Boosting Geranyl Diphosphate Synthesis for Linalool Production in Engineered Yarrowia lipolytica. Appl Biochem Biotechnol, 2023].
Среди микроорганизмов наибольшая продукция линалоола получена при использовании P. ananatis, члена семейства бактерий Enterohacteriaceae, она составила 10,9 г/л. В этом организме для усиления синтеза основных прекурсоров линалоола были сверхэкспрессированы некоторые гены мевалонатного пути, сверхэкспрессирован ген LIS Actinidia arguta [Coutinho, T.A. and S.N. Venter, Pantoea ananatis: an unconventional plant pathogen. Mol Plant Pathol, 2009. 10(3): p. 325-35]. Данный ген кодирует линалоолсинтазу, фермент, ответственный за синтез S-энантиомера линалоола [Hoshino, Y., et al., Stereospecific linalool production utilizing two-phase cultivation system in Pantoea ananatis. Journal of Biotechnology, 2020. 324: p. 21-27]. Однако P. ananatis является патогенным для человека и растений, что сильно ограничивает его применение в биотехнологической промышленности [Coutinho, Т.A. and S.N. Venter, Pantoea ananatis: an unconventional plant pathogen. Mol Plant Pathol, 2009. 10(3): p. 325-35].
В исследовании с использованием Y. lipolytica Xuan Cao с коллегами получили максимальную продукцию линалоола среди дрожжей - 6,96±0,29 мг/л в ферментации в колбах с рабочим объемом 50 мл. Результат был достигнут благодаря гетерологичной экспрессии в родительском штамме гена LIS из A. arguta, а также сверхэкспрессии в нем генов мевалонатного пути и оптимизации процесса ферментации [Cao, X., et al., Enhancing linalool production by engineering oleaginous yeast Yarrowia lipolytica. Bioresource Technology, 2017. 245: p. 1641-1644].
Технической проблемой, на решение которой направлено заявляемое изобретение является расширение арсенала микроорганизмов, продуцирующих линалоол.
Раскрытие сущности изобретения
Техническим результатом является получение штамма дрожжей Yarrowia lipolytica - продуцента линалоола.
Технический результат достигается посредством конструирования рекомбинантного штамма дрожжей Y. lipolytica продуцента линалоола, обладающего следующим генотипом МАТа, Δpex 10 hp4d-YlACBP hp4d-YlZWF1 hp4d-YlACC1 TEFin-YlDGA1 TEFin-ScSUC2 TEFin-YlHXK1 Δku70::Cas9-URA3 IntF1::PTEFin-HMGR1 IntD4::PTEFin-ERG12 IntA2::PTEFin-ERG20F88W-N119W IntB1::PTEFin-ERG20F88W-N119W IntE12::PTEFin-LIS IntE16::PTEFin-LIS IntC14::PTEFin-LIS IntC3::PTEFin-CrGPPS ΔYlDGK1 IntC2::PTEFin-LIS-GSsh-CrGPPS.
Краткое описание чертежей
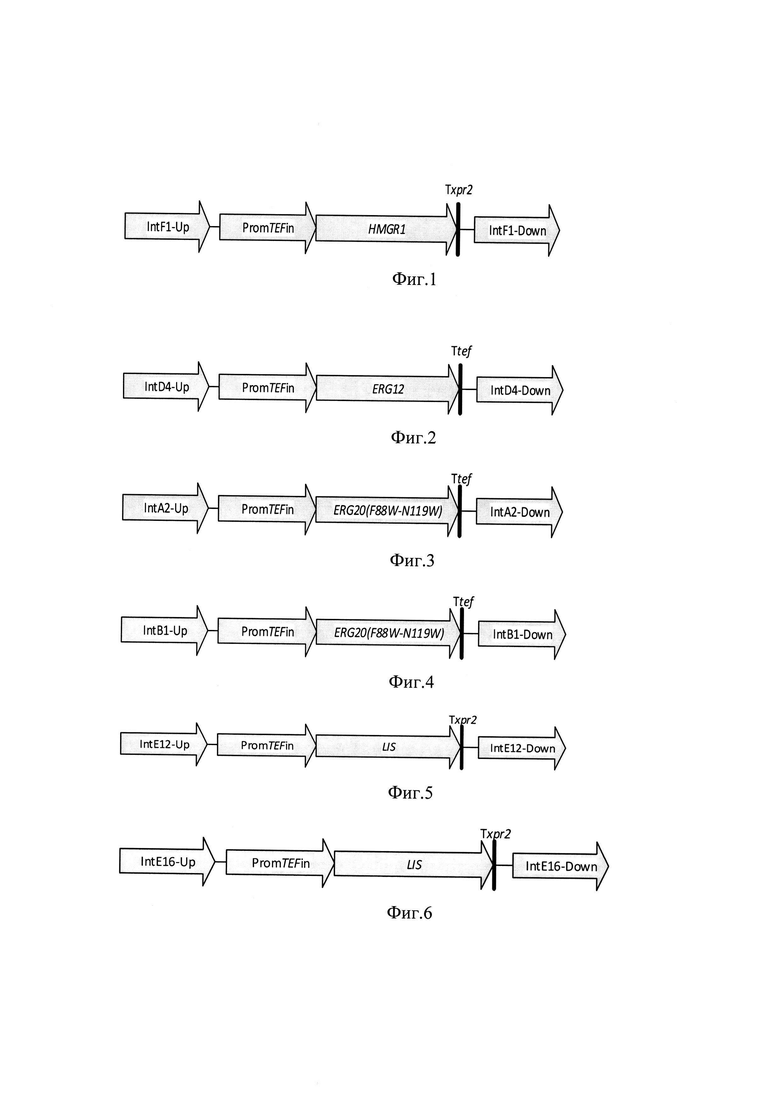
На фиг. 1 показана схема экспрессионной кассеты IntF1-PTEFin-HMGR1.
На фиг. 2 показана схема экспрессионной кассеты IntD4-PTEFin-ERG12.
На фиг. 3 показана схема экспрессионной кассеты IntA2-PTEFin-ERG20F88W-N119W
На фиг. 4 показана схема экспрессионной кассеты IntB1-PTEFin-ERG20F88W-N119W
На фиг. 5 показана схема экспрессионной кассеты IntE12-PTEFin-LIS.
На фиг. 6 показана схема экспрессионной кассеты IntE16-PTEFin-LIS.
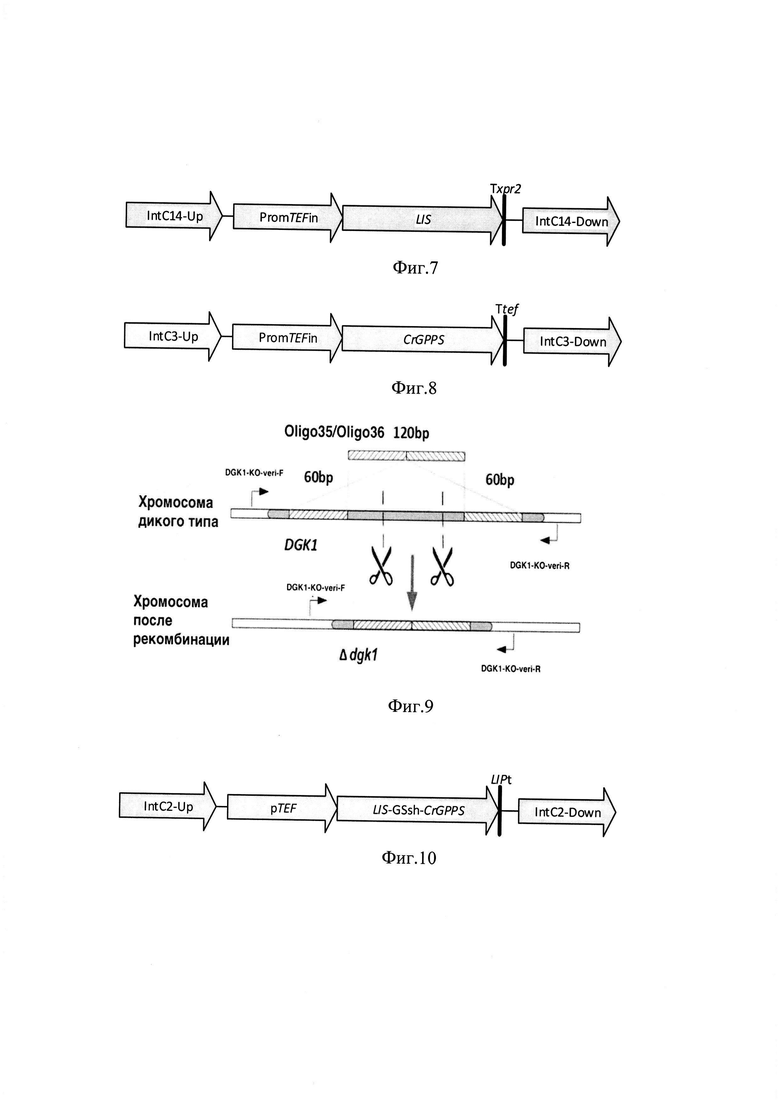
На фиг. 7 показана схема экспрессионной кассеты IntC14-PTEFin-LIS.
На фиг. 8 показана схема экспрессионной кассеты IntC3-PTEFin-CrGPPS.
На фиг. 9 показана схема инактивации гена DGK1 посредством олигонуклеотида Oligo35/Oligo36.
На фиг. 10 показана схема экспрессионной кассеты IntC2-PTEFin-LIS-GSsh-CrGPPS.
Осуществление изобретения
Штамм сконструирован путем следующих последовательных модификаций рекомбинантного штамма Y. lipolytica ВКПМ Y-4620 [Taratynova, М.О., et al., Boosting Geranyl Diphosphate Synthesis for Linalool Production in Engineered Yarrowia lipolytica. Appl Biochem Biotechnol, 2023]:
- последовательная интеграция в геном посредством гомологичной рекомбинации экспрессионных кассеты f1IntF1-HMGR1, f1IntD4-ERG12, f1IntA2-ERG20F88W-N119W, f1IntB1-ERG20F88W-N119W, f1IntE12-LIS, f1IntE16-LIS; f1IntC14-LIS, f1IntC3-CrGPPS;
- инактивация гена DGK1 посредством гомологичной рекомбинации двунитевого олигонуклеотида Oligo35/Oligo36;
интеграция в геном посредством гомологичной рекомбинации экспрессионной кассеты f1IntC2-LIS-GSsh-CrGPPS.
Полученный штамм депонирован в БРЦ ВКПМ НИЦ «Курчатовский институт» (117545 Москва, 1 Дорожный пр-д, д. 1) под регистрационным номером Y. lipolytica ВКПМ Y-5110.
Культурально-морфологические признаки. Суточная культура в жидкой YPD среде (мас. %: дрожжевой экстракт - 1, пептон - 1, глюкоза - 2, вода - остальное) представлена овальными, удлиненно-овальными, округлыми клетками размером 4,6-6,0×4,5-12,5 мкм. Почкование полярное или латеральное, на узком основании. К третьим суткам большинство клеток почкуются и образуют истинный и псевдомицелий.
При росте в жидкой среде YPD при 28°С в течение 24 ч культивирования жидкость мутная, осадок белый, коагуляции не наблюдается, пристеночных пленок не образует.
Клетки хорошо растут на простых питательных средах. Колонии на мальтоагаре (возраст 1 неделя) белого цвета, пастообразные, слегка приподнятые в центре, морщинистые, с фестончатым краем. Штрих на мальтоагаре непрерывный, плоский, блестящий, белого цвета, пастообразный, края ровные, со временем становится складчатым. Спор не образует.
Физиолого-биохимические признаки. Облигатный аэроб. Сахара не сбраживает. Ассимилирует: сахарозу, глюкозу, D-галактозу (медленно), L-сорбозу, D-рибозу, этанол, глицерин, эритрит, адонит, D-маннит, сорбит и молочную, янтарную, лимонную, глюконовую кислоты. Не ассимилирует: мальтозу, лактозу, целлобиозу, трегалозу, мелибиозу, раффинозу, мелицитозу, инулин, крахмал, D-ксилозу, L- и D-арабинозу, раммозу, дульцит, инозит, D-глюкозамин и глюкуроновую, 2-кетоглюконовую, 5-кетоглюконовую кислоты. Не ассимилирует нитраты, жирные кислоты и алканы. Не растет в безвитаминной среде, требует присутствия в среде тиамина, не требует биотина.
Оптимальное значение рН для роста 5,5-7,0. Не растет при 37°С. Максимальная температура роста 35°С. Разжижает желатин. Гидролизует мочевину.
Заявляемый штамм при выращивании в колбах с рабочим объемом 50 мл на среде с сахарозой в качестве единственного источника углерода способен накапливать линалоол в количестве до 109,6 мг/л.
Изобретение проиллюстрировано следующими фигурами графического изображения
Получение и культивирование штамма Y. lipolytica ВКПМ Y-5110 подтверждено следующими примерами конкретного исполнения:
Пример 1. Получение нуклеотидных последовательностей генов CrGPPS и LIS
Здесь и далее все гетерологичные гены кодон-оптимизируют для Y. lipolytica с помощью онлайн программы GenSmart™ [Anderson, S.L., V. Schirf, and L. McAlister-Henn, Effect of AMP on mRNA binding by yeast NAD+ -specific isocitrate dehydrogenase. Biochemistry, 2002. 41(22): p. 7065-73].
Укороченную (начиная со второго метионина (Met 100)) версию гена CrGPPS С. roseus [Fickers, P., et al., Hydrophobic substrate utilisation by the yeast Yarrowia lipolytica, and its potential applications. FEMS Yeast Res, 2005. 5(6-7): p. 527-43] кодон-оптимизируют и синтезируют в виде плазмиды pT-IntC3-CrGPPS (синтез осуществлен компанией «Twist Bioscience Corporation)) (США)). Последовательность кодон-оптимизированного гена CrGPPS приведена в перечне последовательностей под SEQ ID NO:1.
Ген LIS из A. argute (LIS, GenBank ID: GQ33 8153.1) кодон-оптимизируют и синтезируют в виде плазмид pT-f1IntE12-LIS, pT-f1IntE16-LIS и pT-IntC14-LIS в Twist Bioscience Corporation. Последовательность кодон-оптимизированного гена LIS приведена в перечне последовательностей под номером SEQ ID NO:2.
Пример 2. Интеграция гена HMGR1 Y. lipolytica, кодирующего фермент 3-гидрокси-3-метилглютарил-кофермент А редуктазу, в геном штамма-реципиента
Конструируют экспрессионную плазмиду pARS-IntF1-TEFin-HMGR1, содержащую ген HMGR1, для интеграции в локус IntF1 на хромосоме F (положение 3863884-3865837 п.н.) с помощью CRISPR-Cas9 системы [Holkenbrink, С, et al., EasyCloneYALI: CRISPR/Cas9-Based Synthetic Toolbox for Engineering of the Yeast Yarrowia lipolytica. Biotechnol J, 2018. 13(9): p. e1700543]. Плазмиду pARS-IntF1-TEFin-HMGR1 получают на базе вспомогательной плазмиды pARS-IntF1-PromTerm-Cm, которую получают путем сборки 7 фрагментов по методу Гибсона [Gibson, D.G., et al., Enzymatic assembly of DNA molecules up to several hundred kilobases. Nat Methods, 2009. 6(5): p. 343-5].
Фрагмент 1 с фланком гомологии Up к локусу IntF1 (IntF1-Up) амплифицируют с геномной ДНК штамма дикого типа Y. lipolytica W29 [Taratynova, М.О., et al., Boosting Geranyl Diphosphate Synthesis for Linalool Production in Engineered Yarrowia lipolytica. Appl Biochem Biotechnol, 2023] с помощью праймеров:
TTATGCATGCTACTTGGGTGTAATATTGGGATCCGAGCTCATTTA
AATAAGATAATCCAGCGGTGG (IntF1-UP-toARS-SmiI-F);
CAGATATTGTTGTAAATAGAAAATCTGGCTTGTAGGTGGCAGCGC TACTTCTGCAACCTCATGAGAAGCATTTTGATAGTTTAAACCACAAGAA CACGGCTGGG (IntF1-UP-MssI-Eco47III-toTxpr-R).
Фрагмент 2 с терминатором xpr2 (Txpr2) амплифицируют с геномной ДНК штамма Y. lipolytica W29 с помощью праймеров:
ACTATCAAAATGCTTCTCATGAGGTTGCAGAAGTAGCGCTGCCAC CTACAAGCCAGATT (Txpr2-F);
CTGTTTTTTGGGACACAAATGCGCCGCCAACCCGGTCTCTGCGGC CGCTAGGCAATTAACAGATAGTTT (Txpr2-toTEFin-R).
Фрагмент 3 с промотером TEFin (PromTEFin) получают путем обработки вектора pTEFin-uno [Юзбашева, Е.Ю., et al., СПОСОБ ПОВЫШЕНИЯ ПРОДУКЦИИ ИЗОЛИМОННОЙ КИСЛОТЫ У ДРОЖЖЕЙ YARROWIA LIPOLYTICA, ДРОЖЖИ ВИДА YARROWIA LIPOLYTICA, ОБЛАДАЮЩИЕ СПОСОБНОСТЬЮ К ПРОДУКЦИИ ИЗОЛИМОННОЙ КИСЛОТЫ, Н.К. институт", 2020: Россия] эндонуклеазами рестрикции NheI и BpiI и очищают после электрофореза в 1% агарозном геле.
Фрагмент 4 с геном устойчивости к хлорамфениколу (Cm) амплифицируют с плазмиды pMW-att-Cm [Таратынова, М.О., et al., Накопление нейтральных липидов и β-каротина дрожжами Yarrowia lipolytica на среде с сахарозой в качестве источника углерода. Биотехнология, 2021. 37(3): р. 29-41] с помощью праймеров:
CACGTCATCCGACCAGCACTTTTTGCAGTACTAACCGCAGATATG CAGGTGGCTGATGTCCGGCGGTG (Cm-toTEFin-F);
CACAAGACATATCTACAGCAAATGTATAGCAGGTGGCTAACCGTT TTTATCAGGCT (Cm-toEXP1-R).
Фрагмент 5 с терминатором tef (Ttef) амплифицируют с геномной ДНК штамма Y. lipolytica W29 с помощью праймеров:
ACGGGCGCCAAACTCCTTATCCTGCAGGGCTGCTTGTACCTAGTG САА (Ttef-toEXP-F);
AGGCCTAACCTCTGCAACCTGATTTTGATGTTTAATAGTCACGTG ACTGAGTGACAGAGCCCTC (Ttef-R).
Фрагмент 6 с фланком гомологии Down к локусу IntF1 (IntF1-Down) амплифицируют с геномной ДНК штамма Y. lipolytica W29 с помощью праймеров:
ACACCCCACAAAAAGACCCGTGCAGGACATCCTACTGCGTCACG TGACTATTAAACATCAAAATCAGGTTGCAGAGGTTAGGCCTCTGCTTGT TATGGTAGTACTTG (IntF1-Down-Eco147I-F);
CGCCTCTCCCCGCGCGTTGGCCGATTCATTAATGCAGCTGATTTA AATATGGTCTCCCCACCTTG (IntF1-Down-SmiI-to-pMW-R).
Фрагмент 7 получают обработкой вектора pARS-Cre-reverse [Юзбашева, Е.Ю., Юзбашев Т. В., Мостова Е. Б., ДРОЖЖИ РОДА YARROWIA, ОБЛАДАЮЩИЕ СПОСОБНОСТЬЮ ВНУТРИКЛЕТОЧНО НАКАПЛИВАТЬ СЛОЖНЫЕ ЭФИРЫ ЖИРНЫХ КИСЛОТ, И СПОСОБ МИКРОБИОЛОГИЧЕСКОГО СИНТЕЗА ТАКИХ ЭФИРОВ, Н.К. институт", 2015: Россия] эндонуклеазами рестрикции SacI и PvuII, очищают после электрофореза в 1% агарозном геле, обрабатывают Pfu-полимеразой для получения тупых концов.
После реакции сборки по Гибсону, смесь фрагментов трансформируют в Е. coli XL1 (Blue). Плазмидную ДНК, выделенную из полученных клонов, проверяют рестрикционным анализом.
Для получения экспрессионной плазмиды pARS-IntF1-TEFin-HMGR1 фрагмент HMGR1 амплифицируют с геномной ДНК штамма Y. lipolytica W29 с помощью праймеров:
CACGTCATCCGACCAGCACTTTTTGCAGTACTAACCGCAGCTACA AGCAGCTATTGGAAA (HMGR1-to TEFin-F);
AAGAGAATTATCACCGGCAAACTATCTGTTAATTGCCTACCTATG ACCGTATACAAATATTCG (HMGR1-toTxpr2-K).
Вспомогательную плазмиду pARS-IntF1-PromTerm-Cm обрабатывают эндонуклеазой рестрикции AarI, очищают после электрофореза в 1% агарозном геле, обрабатывают щелочной фосфатазой для удаления 5'- и 3'-фосфатных групп с концов ДНК. Полученный фрагмент собирают методом Гибсона с ПЦР-фрагментом HMGR1 с получением плазмиды pARS-IntF1-TEFin-HMGR1.
Вспомогательную плазмиду pCNR-sgIntF1 для осуществления интеграции гена HMGR1 посредством CRISPR-Cas9 системы редактирования генома конструируют с помощью олигонуклеотида
TCGATGGGCCCCCGGTTCGATTCCGGGTCGGCGCAACTGGCAGACAGA TAAGACAGTTTTAGAGCTAGAAATAGCAAGTTAAAATAAGGC (IntF1b-20bp-N) на базе разработанной эписомальной плазмиды pCasNA-RK (Addgene ID: 175708) методом рекомбиниринга [Таратынова, М.О., et al., Накопление нейтральных липидов и β -каротина дрожжами Yarrowia lipolytica на среде с сахарозой в качестве источника углерода. Биотехнология, 2021. 37(3): р. 29-41].
Экспрессионную кассету IntF1-PTEFin-HMGR1 (Фиг. 11) получают путем обработки плазмиды pARS-IntF1-TEFin-HMGR1 эндонуклеазой рестрикции SmiI и используют совместно со вспомогательной кольцевой плазмидой pCNR-sgIntF1 по 1 мкг для ко-трансформации штамма-реципиента Y. lipolytica ВКПМ Y-4620.
Трансформанты отбирают на среде YPSuc Nat (мас. %: дрожжевой экстракт - 1, пептон - 2, сахароза - 1, агар - 1, вода - остальное) с добавлением нурсеотрицина (0,025 мас. %) по устойчивости к антибиотику нурсеотрицину. У трансформантов изолируют геномную ДНК и проводят ПЦР-анализ на правильность интеграции генетической кассеты по праймерам:
AAGTCGCAAACAAAGAGAATTAC (IntF1-chrom-F);
CTATTTGACAAGAATAGCATATCC (IntF1 -chrom-R).
Получают трансформант 2 с кассетой IntF1-PTEFin-HMGR1, интегрированной в локус IntF1 на хромосоме F.
Пример 3. Интеграция гена ERG12 Y. lipolytica, кодирующего мевалонаткиназу
Конструируют экспрессионную плазмиду pARS-IntD4-TEFin-ERG12, содержащую ген ERG12, для интеграции в локус IntD4 на хромосоме D (положение 1038596-1039926 п.н.) с помощью CRISPR-Cas9 системы. Плазмиду pARS-IntD4-TEFin-ERG12 получают на базе вспомогательной плазмиды pARS-IntD4-PromTerm-Cm. Плазмиду pARS-IntD4-PromTerm-Cm конструируют путем сборки 7 фрагментов по методу Гибсона. Все фрагменты получают с помощью ПЦР как описано в примере 2, за исключением фрагментов 1 и 6, получение которых описано ниже.
Фрагмент 1 (IntD4-Up) амплифицируют с геномной ДНК штамма Y. lipolytica W29 с использованием праймеров:
TTATGCATGCTACTTGGGTGTAATATTGGGATCCGAGCTCATTTA AATTTCCCATGGCTGTCTGC (IntD4-UP-toARS-F);
ACTTCTGCAACCTCATGAGAAGCATTTTGATAGTTTAAACTCCGG CCAAGACACATCTG (IntD4-UP-G1-R).
Фрагмент 6 (IntD4-Down) амплифицируют с геномной ДНК штамма Y. lipolytica W29 с использованием праймеров:
GACTATTAAACATCAAAATCAGGTTGCAGAGGTTAGGCCTACCTT TGCTTGTCTTCTGTCA (IntD4-Down-G2-F);
CGCCTCTCCCCGCGCGTTGGCCGATTCATTAATGCAGCTGATTTA AATCTCAGACGGCCCGGAT (IntD4-SmiI-to-vect-R).
Ген ERG12 амплифицируют с геномной ДНК штамма Y. lipolytica W29 с помощью праймеров:
CACGTCATCCGACCAGCACTTTTTGCAGTACTAACCGCAGGACTA CATCATTTCGGCGCCA(ERG12-to-Ptefin-F);
TAATTTTTAACAAACTGGGGTTGCACTAGGTACAAGCAGCCTAAT GGGTCCAGGGACCGAT (ERG12-toTtef-R).
Плазмиду pARS-IntD4-PromTerm-Cm обрабатывают эндонуклеазой рестрикции AarI, очищают после электрофореза в 1% агарозном геле, обрабатывают щелочной фосфатазой для удаления 5'- и 3'-фосфатных групп с концов ДНК. Полученный фрагмент собирают по методу Гибсона с ПЦР-фрагментом ERG12 с получением плазмиды pARS-IntD4-TEFin-ERG12.
Вспомогательную плазмиду pCNR-sgIntD4 для осуществления интеграции гена ERG12 с помощью CRISPR-Cas9 системы конструируют с помощью олигонуклеотида TCGATGGGCCCCCGGTTCGATTCCGGGTCGGCGCAAGTCGTGGTGGATC CTCAGGGTTTTAGAGCTAGAAATAGCAAGTTAAAATAAGGC (IntD4a-20bp-N) на базе разработанной эписомальной плазмиды pCNR-RK методом рекомбиниринга.
Линеаризованный фрагмент ДНК IntD4-PTEFin-ERG12 (Фиг. 2) получают путем обработки плазмиды pARS-IntD4-TEFin-ERG12 эндонуклеазой рестрикции SmiI и используют совместно с вспомогательной кольцевой плазмидой pCNR-sgIntD4 для ко-трансформации трансформанта 2.
Трансформанты отбирают на среде YPSuc Nat по устойчивости к антибиотику нурсеотрицину. У трансформантов изолируют геномную ДНК и проводят ПЦР-анализ на правильность интеграции генетической кассеты по праймерам:
AGCAGGGCTACAGAAGAAAC (IntD4-chr-F);
TATGTCACCGCACCTGTCC (IntD4-chr-R).
Получают трансформант 3 с кассетой IntD4-PTEFin-ERG12, интегрированной в локус IntD4 на хромосоме D.
Пример 4. Интеграция мутантного гена ERG20F88W-N119W Y. lipolytica, кодирующего фарнезил пирофосфат синтазу
Конструируют экспрессионную плазмиду pARS-IntA2-ERG20F88W-N119W, содержащую мутантный вариант гена ERG20 Y. lipolytica с двумя заменами аминокислот F88W и N119W для интеграции в локус IntA2 на хромосоме А (положение 221472-222858 п.н.) с помощью CRISPR-Cas9 системы. Плазмиду pARS-IntA2-ERG20F88W-N119W получают на базе вспомогательной плазмиды pARS-IntA2-Prom-term-Cm. Плазмиду pARS-IntA2-Prom-term-Cm конструируют путем сборки 7 фрагментов по методу Гибсона. Все фрагменты получают с помощью ПЦР как описано в примере 2, за исключением фрагментов 1 и 6, получение которых описано ниже.
Фрагмент 1 (IntA2-Up) амплифицируют с геномной ДНК штамма Y. lipolytica W29 с использованием праймеров:
TTATGCATGCTACTTGGGTGTAATATTGGGATCCGAGCTCATTTA AATTGGGGTCAGATAGTCAC (IntA2-UP-F);
ACTTCTGCAACCTCATGAGAAGCATTTTGATAGTTTAAACGTAAT AAATATTCTATGAAGGTGG (IntA2-UP-R).
фрагмент 6 (IntA2-Down) амплифицируют с геномной ДНК штамма Y. lipolytica W29 с использованием праймеров:
GACTATTAAACATCAAAATCAGGTTGCAGAGGTTAGGCCTAACCC TGGTAAGCACACCC (IntA2-Down-F);
CGCCTCTCCCCGCGCGTTGGCCGATTCATTAATGCAGCTGATTTA AATACAAACCACCTCATTGG (IntA2-Down-R).
Фрагмент from TEFin-ERG20F88W-N119W- to Ttef получают амплификацией с синтезированного в Twist Bioscience Corporation фрагмента ERG20F88W-N119W с использованием пары праймеров:
CACGTCATCCGACCAGCACTTTTTGCAGTACTAACCGCAGTCCAA GGCGAAATTCGAAAGC (ERG20mut-toTEFin-F);
TAATTTTTAACAAACTGGGGTTGCACTAGGTACAAGCAGCCTACT TCTGTCGTTTGTAAATC (ERG20mut-toTtef-R).
Полученный фрагмент fromTEFin-ERG20F88W-N119W-toTtef и плазмиду pARS-IntA2-Prom-term-Cm расщепляют эндонуклеазой рестрикции AarI и собирают методом Гибсона в единую плазмиду pARS-IntA2-ERG20F88W-N119W.
Вспомогательную плазмиду pCNR-sgIntA2 для осуществления интеграции гена ERG20F88W-N119W с помощью CRISPR-Cas9 системы конструируют с помощью олигонуклеотида
TCGATGGGCCCCCGGTTCGATTCCGGGTCGGCGCAACCATTCGAACCGC AAAGCCGTTTTAGAGCTAGAAATAGCAAGTTAAAATAAGGC (IntA2b-20bp-N) на базе разработанной эписомальной плазмиды pCNR-RK методом рекомбиниринга.
Линеаризованный фрагмент IntA2-PTEFin-ERG20F88W-N119W (Фиг. 3) получают путем обработки плазмиды pARS-IntA2-ERG20F88W-N119W эндонуклеазой рестрикции SmiI и ко-трансформируют совместно с вспомогательной плазмидой pCNR-sgIntA2 в трансформант 3.
Трансформанты отбирают на среде YPSuc Nat по устойчивости к антибиотику нурсеотрицину. У трансформантов изолируют геномную ДНК и проводят ПЦР-анализ на правильность интеграции генетической кассеты по праймерам:
GCCCATAATAGACTAGCAAG (IntA2-chrom-F);
GCCAATTATCTCTTATTCAATTC (IntA2-chrom-R).
Получают трансформант 4-1 с кассетой IntA2-ERG20F88W-N119W, интегрированной в локус IntA2 на хромосоме А.
Далее в полученный трансформант 4-1 осуществляют интеграцию второй копии гена ERG20F88W-N119W. Для этого конструируют экспрессионную плазмиду pARS-IntB1-ERG20F88W-N119W, содержащую мутантный вариант гена ERG20 Y. lipolytica с двумя заменами аминокислот F88W и N119W для интеграции в локус IntB1 на хромосоме В (положение 118362-119703 п.н.) с помощью CRISPR-Cas9 системы. Плазмиду pARS-IntB1-ERG20F88W-N119W получают на базе вспомогательной плазмиды pARS-IntB1-Prom-term-Cm. Плазмиду pARS-IntB1-Prom-term-Cm конструируют путем сборки 7 фрагментов по методу Гибсона. Все фрагменты получают с помощью ПЦР как описано в примере 2, за исключением фрагментов 1 и 6, получение которых описано ниже.
Фрагмент 1 (InB1-Up) амплифицируют с геномной ДНК штамма Y. lipolytica W29 с использованием праймеров:
TTATGCATGCTACTTGGGTGTAATATTGGGATCCGAGCTCATTTA AATCAACTAAAAGGAGACCA (IntB1-UP-F);
ACTTCTGCAACCTCATGAGAAGCATTTTGATAGTTTAAACTGCAC ATGAGTAGAAAAATAAAG (IntB1-UP-R).
Фрагмент 6 (IntB1-Down) амплифицируют с геномной ДНК штамма Y. lipolytica W29 с использованием праймеров:
GACTATTAAACATCAAAATCAGGTTGCAGAGGTTAGGCCTCATGT CTTAACCCTTGATTAGA (IntB1-Down-F);
CGCCTCTCCCCGCGCGTTGGCCGATTCATTAATGCAGCTGATTTA AATATGCTCTTGTACTACAATCT (IntB1-Down-R).
Фрагмент fromTEFin-ERG20F88W-N119W-toTtef и плазмиду pARS-IntB1-Prom-term-Cm расщепляют AarI, собирают методом Гибсона в единую плазмиду pARS-IntB1-ERG20F88W-N119W.
Вспомогательную плазмиду pCNR-sgIntB1 для осуществления интеграции гена ERG20F88W-N119W с помощью CRISPR-Cas9 системы конструируют с помощью олигонуклеотида
TCGATGGGCCCCCGGTTCGATTCCGGGTCGGCGCACTTCGGCCAACTAA ATCCTCGTTTTAGAGCTAGAAATAGCAAGTTAAAATAAGGC (IntB1a-20bp-N) на базе разработанной эписомальной плазмиды pCNR-RK методом рекомбиниринга.
Линеаризованный фрагмент IntB1-PTEFin-ERG20F88W-N119W (Фиг. 4) получают путем обработки плазмиды pARS-IntB1-ERG20F88W-N119W эндонуклеазой рестрикции SmiI и ко-трансформируют совместно с вспомогательной плазмидой pCNR-sgIntB1 в трансформант 4-1.
Трансформанты отбирают на среде YPSuc Nat по устойчивости к антибиотику нурсеотрицину. У трансформантов изолируют геномную ДНК и проводят ПЦР-анализ на правильность интеграции генетической кассеты по праймерам:
ACCACCTGTCCTTATCCAAC (IntB1-chrom-F);
GGCATAACCATGGGTTTCAAG (IntB1-chrom-R).
Получают трансформант 4-2 с кассетой IntB1-PTEFin-ERG20F88W-N119W, интегрированной в локус IntB1 на хромосоме В.
Пример 5. Интеграция гена LIS A. arguta, кодирующего линалоолсинтазу
Линеаризованный фрагмент IntE12-PTEFin-LIS (Фиг. 5) получают обработкой плазмиды pT-f1IntE12-LIS эндонуклеазой рестрикции SmiI и ко-трансформируют совместно с вспомогательной плазмидой pCNR-sgIntE12 (Addgene ID: 175636) в трансформант 4-2.
Трансформанты отбирают на среде YPSuc Nat по устойчивости к антибиотику нурсеотрицину. У трансформантов изолируют геномную ДНК и проводят ПЦР-анализ на правильность интеграции генетической кассеты по праймерам:
TTGAATGATGGACTCATAGATG (IntE12-chrom-F);
AAGAGGTCACGTGATGCGG (IntE12-chrom-R).
Получают трансформант 5-1 с кассетой IntE12-PTEFin-LIS, интегрированной в локус IntE12 на хромосоме Е (в положение 2831926-2833196 п.н.).
Далее в полученный трансформант 5-1 осуществляют интеграцию второй копии гена LIS. Для этого линеаризованный фрагмент IntE16-PTEFin-LIS (Фиг. 6) получают обработкой плазмиды pT-f1IntE16-LIS эндонуклеазой рестрикции NotI и ко-трансформируют совместно с вспомогательной плазмидой pCNR-sgIntE16 (Addgene ID: 175637) в трансформант 5-1.
Трансформанты отбирают на среде YPSuc Nat по устойчивости к антибиотику нурсеотрицину. У трансформантов изолируют геномную ДНК и проводят ПЦР-анализ на правильность интеграции генетической кассеты по праймерам:
TCGTGCATTTCGAAACACAAG (IntE16-chrom-F);
ATGTAACACGTTCAGACCAGT (IntE16-chrom-R).
Получают трансформант 5-2 с кассетой IntE16-PTEFin-LIS, интегрированной в локус IntE16 на хромосоме Е (в положение 3975493-3976833 п.н.).
Далее в полученный трансформант 5-2 осуществляют интеграцию третьей копии гена LIS. Для этого линеаризованный фрагмент IntC14-PTEFin-LIS (Фиг. 7) получают обработкой плазмиды pT-f1IntC14-LIS эндонуклеазой рестрикции SmiI и ко-трансформируют совместно с вспомогательной плазмидой pCNR-sgIntC14 (Addgene ID: 175634) в трансформант 5-2.
Трансформанты отбирают на среде YPSuc Nat по устойчивости к антибиотику нурсеотрицину. У трансформантов изолируют геномную ДНК и проводят ПЦР-анализ на правильность интеграции генетической кассеты по праймерам:
AACTTTCTGAAACTTGCTTTCC (IntC14-chrom-F)
AACCAAACACGTGATCAAAGG (IntC14-chrom-R).
Получают трансформант 5-3 с кассетой IntC14-PTEFin-LIS, интегрированной в локус IntC14 на хромосоме С (в положение 3133312-3135244 п.н.).
Пример 6. Интеграция гена CrGPPS С. roseus, кодирующего геранилпирофосфат синтазу
Вспомогательную плазмиду pCNR-sgIntC3 для интеграции гена CrGPPS конструируют, используя олигонуклеотид
TCGATGGGCCCCCGGTTCGATTCCGGGTCGGCGCAACTAACGCAGGAT CAAGAGAGTTTTAGAGCTAGAAATAGCAAGTTAAAATAAGGC (IntC3b-20bp-N), на базе разработанной эписомальной плазмиды pCNR-RK методом рекомбиниринга.
Линеаризованный фрагмент IntC3-PTEFin-CrGPPS (Фиг. 8) получают обработкой плазмиды pT-IntC3-CrGPPS эндонуклеазой рестрикции SmiI и ко-трансформируют совместно с вспомогательной плазмидой pCNR-sgIntC3 в трансформант 5-3.
Трансформанты отбирают на среде YPSuc Nat по устойчивости к антибиотику нурсеотрицину. У трансформантов изолируют геномную ДНК и проводят ПЦР-анализ на правильность интеграции генетической кассеты по праймерам:
AGTCTACTGATTCGGAAAGGC (IntC3-chrom-F);
ACGCTGCTCCTCGTGCAAC (IntC3-chrom-R).
Отбирают трансформант 6 с кассетой IntC3-PTEFin-CrGPPS, интегрированной в локус IntC3 на хромосоме С (в положение 317074-318444 п.н.).
Пример 7. Инактивация гена DGK1, кодирующего диацилглицерол киназу
Ген DGK1 [Zhang, J.L., et al., High production of triterpenoids in. Biotechnol Biofuels, 2020. 13: p. 133] инактивируют в трансформанте 6. Для этого синтезируют однонитевые олигонуклеотиды, комплементарные друг другу
AAACCAATTCTAACACCACCGACACCAACGGCGAAGACCTGTTG GCGTTCTGGCATAATGTCTGAGATCGCATGTCTCTTTCTATACGGCATC ATCATCCCCAAGTACGGCGCTGAAGTC (Oligo35) и
GACTTCAGCGCCGTACTTGGGGATGATGATGCCGTATAGAAAGA GACATGCGATCTCAGACATTATGCCAGAACGCCAACAGGTCTTCGCCG TTGGTGTCGGTGGTGTTAGAATTGGTTT (Oligo36).
Далее путем отжига однонитевых олигонуклеотидов друг на друга получают двунитевой нуклеотид Oligo35/Oligo36 как описано в протоколе (https://www.sigmaaldrich.com/RU/en/technical-documents/protocol/genomics/pcr/annealing-oligos).
Вспомогательные плазмиды pCNR-sgDGK1f и pCNR-sgDGK1d конструируют, используя олигонуклеотиды
TCGATGGGCCCCCGGTTCGATTCCGGGTCGGCGCAACCCTATGGCTGTA CACCCAGTTTTAGAGCTAGAAATAGCAAGTTAAAATAAGGC (DGK1f-20bp-N) и TCGATGGGCCCCCGGTTCGATTCCGGGTCGGCGCACGAACGAGAAGAC CAGAATGGTTTTAGAGCTAGAAATAGCAAGTTAAAATAAGGC (DGK1d-20bp-N), соответственно, на базе разработанной эписомальной плазмиды pCNR-RK методом рекомбиниринга.
Нуклеотидной последовательностью Oligo35/Oligo36 и вспомогательными плазмидами pCNR-sgDGK1f и pCNR-sgDGK1d ко-трансформируют трансформант 6. Схема инактивации гена DGK1, опосредованной CRISPR-Cas9 системой и гомологичной рекомбинацией с олигонуклеотидом Oligo35/Oligo36, представлена на Фиг. 9.
Трансформанты отбирают на среде YPSuc Nat по устойчивости к антибиотику нурсеотрицину. У трансформантов изолируют геномную ДНК и проводят ПЦР-анализ на правильность интеграции нуклеотидной последовательности Oligo35/Oligo36 по праймерам:
ATGTCTGCTGCATCTACTGG (DGK1-KO-veri-F) и
ACTCATTCAGTCTGGTTAGTC (DGK1-KO-veri-R).
Получают трансформант 7 с подтвержденной инактивацией гена DGK1.
Пример 8. Интеграция генов LIS A. argute и CrGPPS, связанных через гибкий глицин-сериновый мостик GSsh
Конструируют интегративную плазмиду pC2US1.1-pTEF-LIS-GSsh-CrGPPS-LIPt, в которой ген LIS из A. argute связан с геном GPPS из С. roseus через гибкий глицин-сериновый мостик GSsh [Woolston, В.М., S. Edgar, and G. Stephanopoulos, Metabolic engineering: past and future. Annu Rev Chem Biomol Eng, 2013. 4: p. 259-88] и находится под контролем промотора pTEF и терминатора LIPt. Последовательность слитых генов LIS и GPPS представлена в перечне последовательностей под номером SEQ ID NO:3. Плазмида предназначена для встраивания в локус IntC2 хромосомы С (в положение 161440-162848 п.н.) с использованием CRISPR-Cas9 системы. Плазмиду получают путем сшивки Golden Gate Assembly (GGA), как описано ранее [Yuzbashev, Т., et al., A DNA assembly toolkit to unlock the CRISPR/Cas9 potential for metabolic engineering. Res Sq, 2023] с использованием эндонуклеазы рестрикции Eco31I из следующих плазмид: pC2US1.1 (Addgene ID 175735), pProC-TEF1 (Addgene ID 175721), pYTK001-Lis-GSsh-CrGPPS-I, pYTK001-CrGPPS-II, pTerC-LIP2 (Addgene ID 175723).
Плазмиду pYTK001-LIS-GSsh-CrGPPS-I получают путем GGA сшивки с использованием эндонуклеазы рестрикции Eco31I плазмиды pYTK001 (Addgene ID 175709) и ПЦР-продукта, содержащего ген LIS. Данный ПЦР-продукт амплифицируют с синтезированного в Twist Bioscience Corporation фрагмента LIS по праймерам:
ATATCGTCTCATCGGGGTCTCAAATGGCCTCTTTCAACCGATTCTG (Lis-I-GGA-F) и TATACGTCTCTGGTCGGTCTCAGGCACTTCTGCGACGACGGATCCGCCT CCGGAAGAGGAGTCGTACAGCA (Lis-I-GGA-R).
Плазмиду pYTK001-CrGPPS-II получают путем GGA сшивки с использованием эндонуклеазы рестрикции Eco31I плазмиды pYTK001 и ПЦР-продукта, содержащего ген CrGPPS. Данный ПЦР-продукт амплифицируют с синтезированного в Twist Bioscience Corporation фрагмента CrGPPS по праймерам:
ATATCGTCTCATCGGGGTCTCATGCCCAAATTAGCCAGCGCAG (CrGPPS-II-F) и
TATACGTCTCTGGTCGGTCTCATAGATTACTTTCGTCGGGTGATGA СТС (CrGPPS-II-R).
Вспомогательную плазмиду pCasNA-IntC2 конструируют, используя олигонуклеотид
TCGATGGGCCCCCGGTTCGATTCCGGGTCGGCGCATCAAAAGTCAGTGT GAGGGGGTTTTAGAGCTAGAAATAGCAAGTTAAAATAAGGC (IntC2-20bp), на базе разработанной эписомальной плазмиды pCNR-RK методом рекомбиниринга.
Линеаризованный фрагмент IntC2-PTEF-LIS-GSsh-CrGPPS (Фиг. 10) получают обработкой плазмиды pC2US1.1-pTEF-LIS-GSsh-CrGPPS-LIPt эндонуклеазой рестрикции MssI и ко-трансформируют совместно со вспомогательной плазмидой pCasNA-IntC2 в трансформант 7.
Трансформанты отбирают на среде YPSuc Nat по устойчивости к антибиотику нурсеотрицину. У трансформантов изолируют геномную ДНК и проводят ПЦР-анализ на правильность интеграции генетической кассеты по праймерам
GTGTGTCCAACCAAAGTGAG (IntC2-chr-F) и
AGAACCGGTCGTTGAGGTC (IntC2-chr-R).
Получают трансформант 8 с подтвержденной интеграцией кассеты в локус IntC2 хромосомы С.Данный трансформант депонируют в БРЦ ВКПМ НИЦ «Курчатовский институт» под регистрационным номером Y. lipolytica ВКПМ Y-5110.
Пример 9. Культивирование штамма Y. lipolytica ВКПМ Y-5110 в колбах.
Культивирование штамма ВКПМ Y-5110 осуществляют в жидкой среде YPSuc9 (мас. %: дрожжевой экстракт - 1, пептон - 1, сахароза - 9, вода - остальное) в колбах (750 мл) с рабочим объемом 50 мл при 28°С при постоянном перемешивании (275 об/мин) в течение 7 суток. На 3 сутки культивирования в колбы добавляют 5 мл изопропилмиристата.
В качестве образца сравнения при культивировании используют трансформант 7, являющийся предшественником штамма ВКПМ Y-5110 и отличающийся от последнего отсутствием кассеты, содержащей гены LIS и CrGPPS, связанные глицин-сериновым мостиком.
После культивирования проводят экстракцию линалоола, для чего в среду культивирования добавляют 2,5 мл изопропилмиристата. Полученный раствор перемешивают (200 об/мин) в течение 1 часа и центрифугируют. Концентрацию линалоола в органической фазе определяют с помощью системы ГХ/МС на приборе «Маэстро» (Interlab) (ГХ Agilent 7820А и МСД 5977, Agilent Technologies). Условия газохроматографического разделения: температура колонки: начальная 70°С в течение 2 мин и нагрев до 260°С со скоростью 15 град/мин; затем нагрев до 320°С со скоростью 30 град/мин и выдержкой при 320°С в течение 1 минуты. Газ-носитель - гелий. Ввод пробы с разделенным на 1/5 газом-носителем. Регистрация масс-спектров в режиме полного сканирования 35-300 а.е. масс. Температура испарителя - 270°С. Температура интерфейса - 280°С, температура квадруполя - 150°С, источника - 230°С. Ионизация электронным ударом, энергия ионизации 70 эВ. Объем вводимого образца: 1 мкл. К 130 мкл гексана добавляли 20 мкл навески изопропилмиристата (разведение в 7,5 раза).
Результаты культивирования штамма Y. lipolytica ВКПМ Y-5110 и его предшественника приведены в Таблице 1.

Полученные результаты показывают, что введение в геном трансформанта 7 кассеты, содержащей гены LIS и CrGPPS, связанные глицин-сериновым мостиком, позволяет получить повышение продукции линалоола в 3,3 раза.
Таким образом, продукция линалоола, получаемая при культивировании штамма Y. lipolytica ВКПМ Y-5110, в 15,7 раза превосходит результат, ранее описанный на дрожжах Y. lipolytica (6,96 мг/л) [Cao, X., et al., Enhancing linalool production by engineering oleaginous yeast Yarrowia lipolytica. Bioresource Technology, 2017.245: p. 1641-1644].

--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing originalFreeTextLanguageCode="ru"
nonEnglishFreeTextLanguageCode="ru" dtdVersion="V1_3"
fileName="Strain Yarrowia lipolytica is a producer of linalool.xml"
softwareName="WIPO Sequence" softwareVersion="2.1.2"
productionDate="2023-09-08">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText></ApplicationNumberText>
<FilingDate>2023-09-07</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>532666768</ApplicantFileReference>
<ApplicantName languageCode="ru">Национальный исследовательский
центр "Курчатовский институт"</ApplicantName>
<ApplicantNameLatin>National Research Center "Kurchatov
Institute"</ApplicantNameLatin>
<InventionTitle languageCode="ru">Штамм дрожжей Yarrowia lipolytica
– продуцент линалоола</InventionTitle>
<SequenceTotalQuantity>3</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>963</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..963</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Yarrowia lipolytica</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtcgtcgcagaagtccccaaattagccagcgcagcagaatactttttca
agctgggcgttgagggcaagcgattccgacccaccgtccttctgctcatggccactgccatcgacgcccc
catcagtcgaactccccccgacacttcccttgacaccctgtccaccgagctgcgactccgacagcagtcc
attgccgagattactgagatgattcatgtggcttctctgttgcatgacgacgtgttggacgacgctgaga
ctcgacggggcattggctctctcaacttcgttatgggcaacaagctcgctgtcctcgcgggcgacttcct
gctgtctcgggcgtgtgttgcgctggcgtcgctaaagaacaccgaggttgttagcctactagcgaccgtt
gtcgaacatctggtcaccggcgaaaccatgcagatgaccaccactagcgaccagcggtgttccatggagt
actacatgcagaaaacctactacaagacagcgtccctgatttccaactcctgtaaggctattgctctgct
cgctggccagacctccgaggtcgccatgctcgcctacgagtacggcaagaacctcggcctcgccttccag
ctcattgacgacgtcctcgacttcaccggaacttcggcctcgctcggaaagggatccctctccgacattc
ggcatggaattgtcactgctcccattctgttcgctattgaggagttccccgagctgcgagctgtggtgga
cgagggattcgagaacccctacaacgtggacctggccctgcattaccttggaaagtcccgaggaatccag
cgaacacgagagcttgccatcaagcatgccaacctggcctctgacgccatcgactctctgcccgtgaccg
acgatgagcatgtcctccgatctcgacgagccctcgtcgagctcacccagcgagtcatcacccgacgaaa
gtag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>1722</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..1722</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q5">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Yarrowia lipolytica</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcctctttcaaccgattctgcgtgtcctccctgctggctcccaacaact
ctccccagatctccaacgctccccgatctaccgctgtgccctccatgcccaccacccagaagtggtctat
taccgaggacctggccttcatctctaacccctccaagcagcacaaccaccagaccggctaccgaattttc
tccgacgagttctacctgaagcacgagaacaagctcaaggacgtccgacgagccctgcgagaggtggagg
agactcccctggagggcctcgtcatgattgacaccctgcagcgactcggtatcgactaccacttccaggg
agagattggagctctgctccagaagcagcagcgaatctctacctgcgactaccccgagcacgacctgttc
gaggtgtccacccgattccgactgctccgacaggagggtcacaacgtccccgccgacgtgttcaacaact
tccgagacaaggagggacgattcaagtctgagctgtcccgagacattcgaggtctgatgtccctctacga
ggcttcccagctgtccattcagggcgaggacatcctcgaccaggccgctgacttctcctctcagctgctc
tctggttgggctaccaacctggaccaccaccaggcccgactggtccgaaacgctctcacccacccctacc
acaagtccctcgccaccttcatggctcgaaacttcaactacgactgtaagggccagaacggttgggtcaa
caacctccaggagctcgccaagatggacctgaccatggtgcagtctatgcaccagaaggaggtcctccag
gtgtcccagtggtggaagggacgaggtctggctaacgagctgaagctcgtgcgaaaccagcccctgaagt
ggtacatgtggcctatggccgctctcaccgaccctcgattctctgaggagcgagtcgagctgaccaagcc
catctccttcatctacatcattgacgacatcttcgacgtgtacggaaccctcgaggagctgaccctcttc
accgacgccgtcaaccgatgggagctgaccgctgtggagcagctccctgactacatgaagatctgcttca
aggccctgtacgacatcaccaacgagattgcttacaagatctacaagaagcacggacgaaaccccattga
ctctctccgacgaacctgggcctccctgtgtaacgctttcctcgaggaggccaagtggttcgcttctggc
aacctgcccaaggccgaggagtacctcaagaacggtatcatttcctctggaatgcacgtcgtgaccgtcc
acatgttcttcctgctcggcggttgtttcaccgaggagtccgtcaacctggtggacgagcacgccggtat
cacctcctctatcgctaccattctgcgactctctgacgacctcggatccgccaaggacgaggaccaggac
ggatacgacggctcttacctggagtgctacctcaaggaccacaagggttcctctgtcgagaacgcccgag
aggaggtcatccgaatgatttccgacgcttggaagcgactgaacgaggagtgcctgtttcccaacccctt
ctctgccaccttccgaaagggatccctgaacatcgctcgaatggtgcccctcatgtactcttacgacgac
aaccacaacctgcccattctcgaggagcacatgaaaaccatgctgtacgactcctcttcctaa</INSDS
eq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>2694</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..2694</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Yarrowia lipolytica</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcctctttcaaccgattctgcgtgtcctccctgctggctcccaacaact
ctccccagatctccaacgctccccgatctaccgctgtgccctccatgcccaccacccagaagtggtctat
taccgaggacctggccttcatctctaacccctccaagcagcacaaccaccagaccggctaccgaattttc
tccgacgagttctacctgaagcacgagaacaagctcaaggacgtccgacgagccctgcgagaggtggagg
agactcccctggagggcctcgtcatgattgacaccctgcagcgactcggtatcgactaccacttccaggg
agagattggagctctgctccagaagcagcagcgaatctctacctgcgactaccccgagcacgacctgttc
gaggtgtccacccgattccgactgctccgacaggagggtcacaacgtccccgccgacgtgttcaacaact
tccgagacaaggagggacgattcaagtctgagctgtcccgagacattcgaggtctgatgtccctctacga
ggcttcccagctgtccattcagggcgaggacatcctcgaccaggccgctgacttctcctctcagctgctc
tctggttgggctaccaacctggaccaccaccaggcccgactggtccgaaacgctctcacccacccctacc
acaagtccctcgccaccttcatggctcgaaacttcaactacgactgtaagggccagaacggttgggtcaa
caacctccaggagctcgccaagatggacctgaccatggtgcagtctatgcaccagaaggaggtcctccag
gtgtcccagtggtggaagggacgaggtctggctaacgagctgaagctcgtgcgaaaccagcccctgaagt
ggtacatgtggcctatggccgctctcaccgaccctcgattctctgaggagcgagtcgagctgaccaagcc
catctccttcatctacatcattgacgacatcttcgacgtgtacggaaccctcgaggagctgaccctcttc
accgacgccgtcaaccgatgggagctgaccgctgtggagcagctccctgactacatgaagatctgcttca
aggccctgtacgacatcaccaacgagattgcttacaagatctacaagaagcacggacgaaaccccattga
ctctctccgacgaacctgggcctccctgtgtaacgctttcctcgaggaggccaagtggttcgcttctggc
aacctgcccaaggccgaggagtacctcaagaacggtatcatttcctctggaatgcacgtcgtgaccgtcc
acatgttcttcctgctcggcggttgtttcaccgaggagtccgtcaacctggtggacgagcacgccggtat
cacctcctctatcgctaccattctgcgactctctgacgacctcggatccgccaaggacgaggaccaggac
ggatacgacggctcttacctggagtgctacctcaaggaccacaagggttcctctgtcgagaacgcccgag
aggaggtcatccgaatgatttccgacgcttggaagcgactgaacgaggagtgcctgtttcccaacccctt
ctctgccaccttccgaaagggatccctgaacatcgctcgaatggtgcccctcatgtactcttacgacgac
aaccacaacctgcccattctcgaggagcacatgaaaaccatgctgtacgactcctcttccggaggcggat
ccgtcgtcgcagaagtgcccaaattagccagcgcagcagaatactttttcaagctgggcgttgagggcaa
gcgattccgacccaccgtccttctgctcatggccactgccatcgacgcccccatcagtcgaactcccccc
gacacttcccttgacaccctgtccaccgagctgcgactccgacagcagtccattgccgagattactgaga
tgattcatgtggcttctctgttgcatgacgacgtgttggacgacgctgagactcgacggggcattggctc
tctcaacttcgttatgggcaacaagctcgctgtcctcgcgggcgacttcctgctgtctcgggcgtgtgtt
gcgctggcgtcgctaaagaacaccgaggttgttagcctactagcgaccgttgtcgaacatctggtcaccg
gcgaaaccatgcagatgaccaccactagcgaccagcggtgttccatggagtactacatgcagaaaaccta
ctacaagacagcgtccctgatttccaactcctgtaaggctattgctctgctcgctggccagacctccgag
gtcgccatgctcgcctacgagtacggcaagaacctcggcctcgccttccagctcattgacgacgtcctcg
acttcaccggaacttcggcctcgctcggaaagggatccctctccgacattcggcatggaattgtcactgc
tcccattctgttcgctattgaggagttccccgagctgcgagctgtggtggacgagggattcgagaacccc
tacaacgtggacctggccctgcattaccttggaaagtcccgaggaatccagcgaacacgagagcttgcca
tcaagcatgccaacctggcctctgacgccatcgactctctgcccgtgaccgacgatgagcatgtcctccg
atctcgacgagccctcgtcgagctcacccagcgagtcatcacccgacgaaagtaa</INSDSeq_seque
nce>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Трансформант дрожжей Yarrowia lipolytica, продуцирующий линалоол | 2022 |
|
RU2809554C1 |
| Модифицированная нуклеотидная последовательность, кодирующая полипептид, обладающий активностью фитоен синтазы и геранилгеранилпирофосфат синтазы (варианты), ее использование при конструировании трансформантов дрожжей Yarrowia lipolytica, продуцирующих каротиноиды | 2021 |
|
RU2794980C1 |
| Трансформант дрожжей Yarrowia lipolytica, продуцирующий кантаксантин | 2023 |
|
RU2827562C1 |
| Способ повышения продукции изолимонной кислоты у дрожжей Yarrowia lipolytica, дрожжи вида Yarrowia lipolytica, обладающие способностью к продукции изолимонной кислоты | 2018 |
|
RU2713124C2 |
| Промотор для экспрессии генов и его использование при конструировании трансформанта дрожжей Yarrowia lipolytica, продуцирующего β-каротин | 2022 |
|
RU2819535C1 |
| ДРОЖЖИ РОДА YARROWIA, ОБЛАДАЮЩИЕ СПОСОБНОСТЬЮ ВНУТРИКЛЕТОЧНО НАКАПЛИВАТЬ СЛОЖНЫЕ ЭФИРЫ ЖИРНЫХ КИСЛОТ, И СПОСОБ МИКРОБИОЛОГИЧЕСКОГО СИНТЕЗА ТАКИХ ЭФИРОВ | 2013 |
|
RU2539744C1 |
| РЕКОМБИНАНТНЫЙ ШТАММ ДРОЖЖЕЙ YARROWIA-LIPOLYTICA - ПРОДУЦЕНТ ЛИПАЗЫ | 2011 |
|
RU2451075C1 |
| ШТАММ ДРОЖЖЕЙ Yarrowia lipolytica - ПРОДУЦЕНТ ЛИПАЗЫ | 2007 |
|
RU2355754C1 |
| Штамм дрожжей Yarrowia lipolytica, продуцирующий изолимонную кислоту | 2021 |
|
RU2757603C1 |
| ИНТЕГРАТИВНЫЙ ВЕКТОР Random-URA3-RPT ДЛЯ ПОСЛЕДОВАТЕЛЬНОГО ВВЕДЕНИЯ МНОЖЕСТВЕННЫХ КОПИЙ ГЕНЕТИЧЕСКИХ ЭЛЕМЕНТОВ В ДРОЖЖИ Yarrowia lipolytica | 2006 |
|
RU2376376C2 |
Изобретение относится к биотехнологии. Предложен штамм дрожжей Y. lipolytica ВКПМ Y-5110, имеющий генотип МАТа, Δpex10 hp4d-YlACBP hp4d-YlZWF1 hp4d-YlACC1 TEFin-YlDGA1 TEFin-ScSUC2 TEFin-YlHXK1 Δku70::Cas9-URA3 IntF1::PTEFin-HMGR1 IntD4::PTEFin-ERG12 IntA2::PTEFin-ERG20F88W-N119W IntB1::PTEFin-ERG20F88W-N119W IntE12::PTEFin-LIS IntE16::PTEFin-LIS IntC14::PTEFin-LIS IntC3::PTEFin-CrGPPS ΔYlDGK1 IntC2::PTEF-LZS-GSsh-CrGPPS и являющийся продуцентом линалоола. Изобретение обеспечивает повышение продукции линалоола. 10 ил., 1 табл., 9 пр.
Штамм дрожжей Y. lipolytica ВКПМ Y-5110, имеющий генотип МАТа, Δpex10 hp4d-YlACBP hp4d-YlZWF1 hp4d-YlACC1 TEFin-YlDGA1 TEFin-ScSUC2 TEFin-YlHXK1 Δku70::Cas9-URA3 IntF1::PTEFin-HMGR1 IntD4::PTEFin-ERG12 IntA2::PTEFin-ERG20F88W-N119W IntB1::PTEFin-ERG20F88W-N119W IntE12::PTEFin-LIS IntE16::PTEFin-LIS IntC14::PTEFin-LIS IntC3::PTEFin-CrGPPS ΔYlDGK1 IntC2::PTEF-LIS-GSsh-CrGPPS, - продуцент линалоола.
| Трансформант дрожжей Yarrowia lipolytica, продуцирующий линалоол | 2022 |
|
RU2809554C1 |
| CAO X | |||
| ET AL | |||
| Enhancing linalool production by engineering oleaginous yeast Yarrowia lipolytica | |||
| Bioresour Technol | |||
| Автомобиль-сани, движущиеся на полозьях посредством устанавливающихся по высоте колес с шинами | 1924 |
|
SU2017A1 |
| Печь-кухня, могущая работать, как самостоятельно, так и в комбинации с разного рода нагревательными приборами | 1921 |
|
SU10A1 |
| CN 108998383 A, 14.12.2018 | |||
| CN 110106209 A, 09.08.2019. | |||
Авторы
Даты
2024-05-21—Публикация
2023-12-19—Подача