Область техники
Настоящее изобретение относится к молекулярной биологии и медицине, а конкретно к способу применения коротких прокариотических аргонавтов для детекции специфических нуклеиновых кислот. Детекция специфических нуклеиновых кислот применяется в тест-системах для определения инфекции человека и животных к различным возбудителям (вирусы гепатита B или C, ВИЧ, SARS-CoV-2 и т.д.).
Изобретение создано в рамках реализации гранта РНФ №22-14-00182 «Программируемые ДНК-зависимые РНК-нуклеазы - защитная система бактерий и инструмент для регуляции экспрессии генов»
Уровень техники
В последние годы ведется разработка тест-систем, основанных на способности некоторых белков использовать нуклеиновые кислоты, т.н. гиды, для узнавания специфических последовательностей ДНК/РНК, т.н. мишени. Такие системы разрабатывают, например, на основе белков системы CRISPR, которые, используя РНК-гиды специальной структуры, узнают комплементарные гидам ДНК или РНК мишени. Связывание гида и мишени стимулирует ДНКазную либо РНКазную активность эффекторного белка комплекса Cas. В зависимости от активности Cas в качестве репортера могут быть использованы нуклеиновые кислоты разной природы. В системах на основе Cas9 расщепляется непосредственно узнаваемая ДНК (Azhar et al., 2020). В системах на основе Cas12 или Cas13 расщепляется ДНК либо РНК, не относящаяся к мишени, так называемая коллатеральная (Ding et al., 2020, 2021; Fozouni et al., 2021). Будучи меченной с одной стороны флуорофором, а с другой гасителем, при расщеплении коллатеральная ДНК либо РНК будет давать флуоресцентный сигнал. Однако, системы на базе CRISPR белков неудобны тем, что белки Cas требуют для активности длинные гиды с определенной структурой и специальную последовательность PAM, которая неравномерно распределена в целевых последовательностях. Кроме того, PAM последовательности могут быть настолько длинными и сложными, что это затрудняет их применение.
Совсем недавно начались исследования похожих систем, но на основе белков аргонавтов прокариот. Аргонавты прокариот используют короткие нуклеиновые кислоты длиной 15 -25 нк в качестве гида и могут узнавать разнообразные мишени. Эти белки можно разделить на несколько больших групп по составу доменов и активности PIWI домена - длинные активные, длинные не активные и короткие не активные (Swarts et al., 2014; Ryazansky, Kulbachinskiy & Aravin, 2018). Тест-системы разрабатываются на основе как длинных, так и коротких аргонавтов.
Так, длинный активный аргонавт археи Pyrococcus furiosus применяется в тест-системе для детекции целевой ДНК (He et al., 2019). Однако эта система является очень сложной и требует использования как набора из трех гидов, а также и специальных репортерных молекул ДНК для детекции каждой целевой ДНК отдельно. Кроме того, оптимальной температурой активности аргонавта из Pyrococcus furiosu является 95°С, что значительно затрудняет его применение в полевых условиях (Wang et al., 2021).
Короткие белки-аргонавты сами не обладают активностью, так как не содержат каталитического центра в домене PIWI нужных остатков, но белки, закодированные рядом с этими аргонавтами в геноме, могут иметь различные активности (Swarts et al., 2014; Ryazansky et al., 2018). Известно несколько типов таких белков - с предсказанной НАДазной активностью (способностью расщеплять НАД), способностью формировать трансмембранные каналы и предсказанной нуклеазной активностью (Kim, Jung & Lim, 2020; Koopal et al., 2022; Zaremba et al., 2022; Zeng et al., 2022; Song et al., 2023).
На данной момент опубликованы работы о некоторых белках с НАДазной активностью. Тест-системы, которые предлагаются к применению в этих работах, являются наиболее близкими аналогами предлагаемой в данной изобретении тест-системы. Показано, что аргонавт и его белок-партнер с НАДазной активностью формируют стабильный комплекс, НАДазная активность которого появляется при связывании гида и мишени Koopal et al., 2022; Zaremba et al., 2022). Так как НАДазная активность появляется только при узнавании комплексом аргонавта и гида целевой мишени, то появление этой активности можно использовать, как сенсор на наличие мишени в реакционной среде (Koopal et al., 2022; Zaremba et al., 2022). Показана принципиальная возможность использования этого белка и флуоресцентного аналога НАД в качестве субстрата в сочетании с плашечным ридером в качестве детектора для определения целевых ДНК. Однако использование аналога НАД сильно повышает стоимость такой системы. Кроме того, применение флуоресцентного аналога НАД значительно ограничивает способы детекции сигнала от такой системы, так как, в данном случае применим только один канал детекции, а также для детекции необходим планшетный ридер, которым не всегда оборудованы диагностические лаборатории.
Azhar, M., Phutela, R., Hussain Ansari, A., Sinha, D., Sharma, N., Kumar, M., Aich, M., Sharma, S., Rauthan, R., Singhal, K., Lad, H., Patra, P.K., Makharia, G., Chandak, G.R., Chakraborty, D. & Maiti, S. (2020). Rapid, field-deployable nucleobase detection and identification using FnCas9.
Ding, X., Yin, K., Li, Z., Lalla, R.V., Ballesteros, E., Sfeir, M.M. & Liu, C. (2020). Ultrasensitive and visual detection of SARS-CoV-2 using all-in-one dual CRISPR-Cas12a assay. Nat. Commun. 2020 111 11, 1-10.
Ding, X., Yin, K., Li, Z., Sfeir, M.M. & Liu, C. (2021). Sensitive quantitative detection of SARS-CoV-2 in clinical samples using digital warm-start CRISPR assay. Biosens. Bioelectron. 184.
Fozouni, P., Son, S., Díaz de León Derby, M., Knott, G.J., Gray, C.N., D’Ambrosio, M.V., Zhao, C., Switz, N.A., Kumar, G.R., Stephens, S.I., Boehm, D., Tsou, C.L., Shu, J., Bhuiya, A., Armstrong, M., Harris, A.R., Chen, P.Y., Osterloh, J.M., Meyer-Franke, A., Joehnk, B., Walcott, K., Sil, A., Langelier, C., Pollard, K.S., Crawford, E.D., Puschnik, A.S., Phelps, M., Kistler, A., DeRisi, J.L., Doudna, J.A., Fletcher, D.A. & Ott, M. (2021). Amplification-free detection of SARS-CoV-2 with CRISPR-Cas13a and mobile phone microscopy. Cell 184, 323-333.e9.
He, R., Wang, L., Wang, F., Li, W., Liu, Y., Li, A., Wang, Y., Mao, W., Zhai, C. & Ma, L. (2019). Pyrococcus furiosusArgonaute-mediated nucleic acid detection. Chem. Commun. 55, 13219-13222.
Kim, S.Y., Jung, Y. & Lim, D. (2020). Argonaute system of Kordia jejudonensis is a heterodimeric nucleic acid-guided nuclease. Biochem. Biophys. Res. Commun. 525, 755-758.
Koopal, B., Potocnik, A., Mutte, S.K., Vervoort, J.J.M.M., Brouns, S.J.J.J., Swarts, D.C., Aparicio-Maldonado, C., Lindhoud, S., Vervoort, J.J.M.M., Brouns, S.J.J.J. & Swarts, D.C. (2022). Short prokaryotic Argonaute systems trigger cell death upon detection of invading DNA. Cell 0, 1-16.
Ryazansky, S., Kulbachinskiy, A. & Aravin, A.A. (2018). The expanded universe of prokaryotic argonaute proteins. mBio 9, 1-20.
Song, X., Lei, S., Liu, S., Liu, Y., Fu, P., Zeng, Z., Yang, K., Chen, Y., Li, M., She, Q. & Han, W. (2023). Long-B prokaryotic Argonaute systems employ various effectors to confer immunity via abortive infection. bioRxiv 2023.03.09.531850.
Swarts, D.C., Makarova, K., Wang, Y., Nakanishi, K., Ketting, R.F., Koonin, E.V., Patel, D.J., Van Der Oost, J., Struct, N. & Biol, M. (2014). The evolutionary journey of Argonaute proteins HHS Public Access Author manuscript. Nat Struct Mol Biol 21, 743-753.
Wang, F., Yang, J., He, R., Yu, X., Chen, S., Liu, Y., Wang, L., Li, A., Liu, L., Zhai, C. & Ma, L. (2021). PfAgo-based detection of SARS-CoV-2. Biosens. Bioelectron. 177, 112932.
Zaremba, M., Dakineviciene, D., Golovinas, E., Zagorskaitė, E., Stankunas, E., Lopatina, A., Sorek, R., Manakova, E., Ruksenaite, A., Silanskas, A., Asmontas, S., Grybauskas, A., Tylenyte, U., Jurgelaitis, E., Grigaitis, R., Timinskas, K., Venclovas, Č. & Siksnys, V. (2022). Short prokaryotic Argonautes provide defence against incoming mobile genetic elements through NAD+ depletion. Nat. Microbiol. 7, 1857-1869.
Zeng, Z., Chen, Y., Pinilla-Redondo, R., Shah, S.A., Zhao, F., Wang, C., Hu, Z., Wu, C., Zhang, C., Whitaker, R.J., She, Q. & Han, W. (2022). A short prokaryotic Argonaute activates membrane effector to confer antiviral defense. Cell Host Microbe 30, 930-943.e6.
Краткое описание изобретения
Сущностью настоящего изобретения является создание новых универсальных и удобных тест-систем для детекции специфических ДНК или РНК, применимых в полевых условиях.
Для решения этой задачи настоящее изобретение предполагает использование для детекции целевых ДНК или РНК комплекс короткого белка-аргонавта и его белка-партнера с нуклеазной активностью, имеющих последовательности SEQ ID NO: 1 и SEQ ID NO: 2, соответственно (последовательности белков из бактерии Novosphingopyxis baekryungensis, белок-аргонавт NbaAgo и его белок-партнер DREN (DNA RNA endonuclease)). Кроме непосредственно белков NbaAgo и DREN, с этой же целью могут быть использованы комплексы гомологичных белков, с уровнем идентичности не менее 20% SEQ ID NO: 1 и SEQ ID NO: 2.
В некоторых вариантах осуществления описана тест-система для детекции целевых молекул нуклеиновых кислот на основе комплекса, состоящего из: 1) белка короткого аргонавта из бактерии Novosphingopyxis baekryungensis (NbaAgo), имеющего аминокислотную последовательность по крайней мере на 20% идентичную последовательности SEQ ID NO: 1; 2) его белка-партнера DREN из бактерии Novosphingopyxis baekryungensis, имеющего аминокислотную последовательность по крайней мере на 20% идентичную последовательности SEQ ID NO: 2; а также гида (направляющей РНК), мишени (молекулы нуклеиновой кислоты-мишени) и универсального флуоресцентного репортера - флуоресцентной репортерной нуклеиновой кислоты, причем флуоресцентная репортерная нуклеиновая кислота имеет флуоресцентную группу и гасящую группу.
В некоторых вариантах осуществления предполагается тест-система, в которой в качестве направляющей РНК используется последовательность SEQ ID NO: 3, в качестве мишени - последовательность SEQ ID NO: 4.
В некоторых вариантах осуществления в качестве флуоресцентной репортерной нуклеиновой кислоты используется оцДНК.
В некоторых вариантах осуществления в качестве флуоресцентной репортерной нуклеиновой кислоты используется дцДНК.
В некоторых вариантах осуществления в качестве флуоресцентной репортерной нуклеиновой кислоты используются последовательности SEQ ID NO: 5 (BHQ2-GTCACAGAGATACTACGTGTGCGACTGCTCAG-ROX), SEQ ID NO: 6 (GTCACAGAGATACTACGTGTGCGACTGCTCAG-ROX) или SEQ ID NO: 7 (CTGAGCAGTCGCACATGTAGTATCTCTGTGAC-BHQ2).
В некоторых вариантах осуществления предполагается применение для детекции целевых ДНК или РНК комплекса, состоящего из: 1) белка короткого аргонавта из бактерии Novosphingopyxis baekryungensis (NbaAgo), имеющего аминокислотную последовательность по крайней мере на 20% идентичную последовательности SEQ ID NO: 1; 2) его белка-партнера DREN из бактерии Novosphingopyxis baekryungensis, имеющего аминокислотную последовательность по крайней мере на 20% идентичную последовательности SEQ ID NO: 2; а также гида (направляющей РНК), мишени (молекулы нуклеиновой кислоты-мишени) и универсального флуоресцентного репортера - флуоресцентной репортерной нуклеиновой кислоты, причем флуоресцентная репортерная нуклеиновая кислота имеет флуоресцентную группу и гасящую группу.
В некоторых вариантах осуществления предполагается применение комплекса, где в качестве направляющей РНК используется последовательность SEQ ID NO: 3, а в качестве мишени - последовательность SEQ ID NO: 4.
В некоторых вариантах осуществления предполагается применение комплекса, где в качестве флуоресцентной репортерной нуклеиновой кислоты используется оцДНК или дцДНК.
В некоторых вариантах осуществления предполагается применение комплекса, где в качестве флуоресцентной репортерной нуклеиновой кислоты - последовательности SEQ ID NO: 5, SEQ ID NO: 6 или SEQ ID NO: 7.
В некоторых вариантах осуществления предполагается применение комплекса при температуре от 20°С до 40°С.
В некоторых вариантах осуществления предполагается способ определения наличия в образце молекулы нуклеиновой кислоты-мишени, включающий следующие этапы: (a) предоставление тест-системы для детекции по п.1 для обнаружения молекул нуклеиновой кислоты-мишени; (b) взаимодействие тест-системы для детекции по п.1 с подлежащим обнаружению образцом при температуре от 20°С до 40°С, в результате чего образуется реакционный раствор; (c) детекция флуоресценции в реакционном растворе; при этом, если в реакционном растворе определяется значение сигнала флуоресценции, это означает, что целевая молекула нуклеиновой кислоты существует в образце; а если значение сигнала флуоресценции не определяется в реакционном растворе, это означает, что мишень не существует в молекулах нуклеиновой кислоты образца.
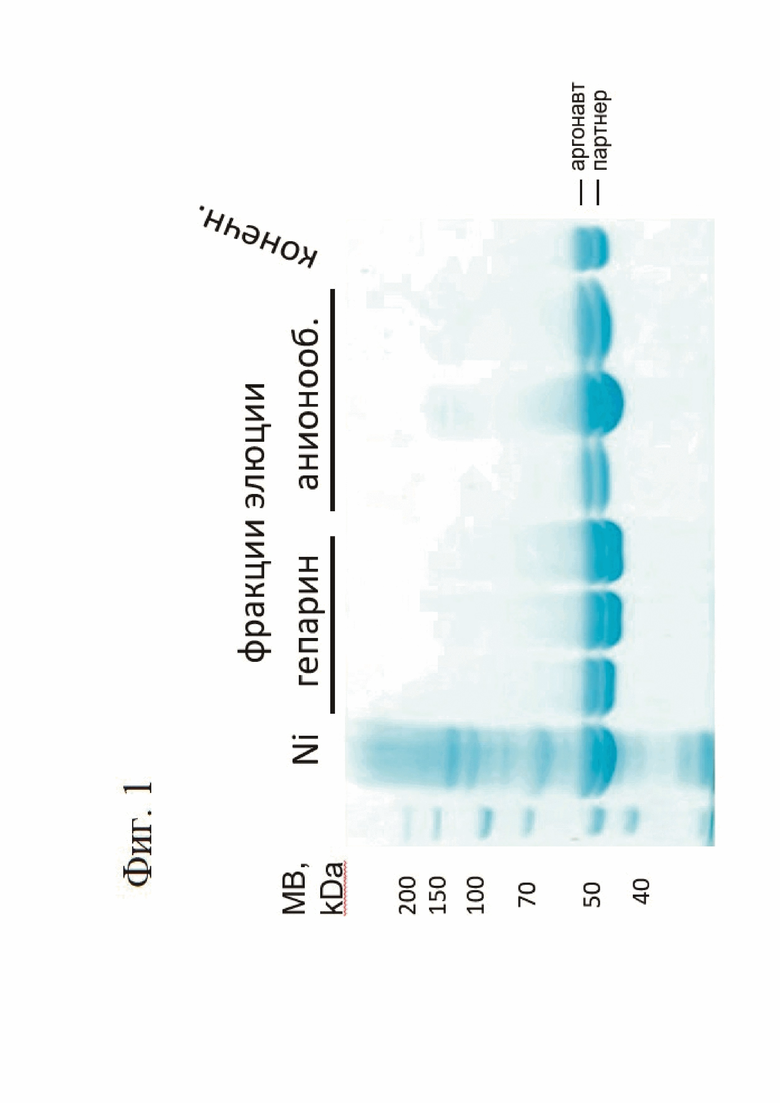
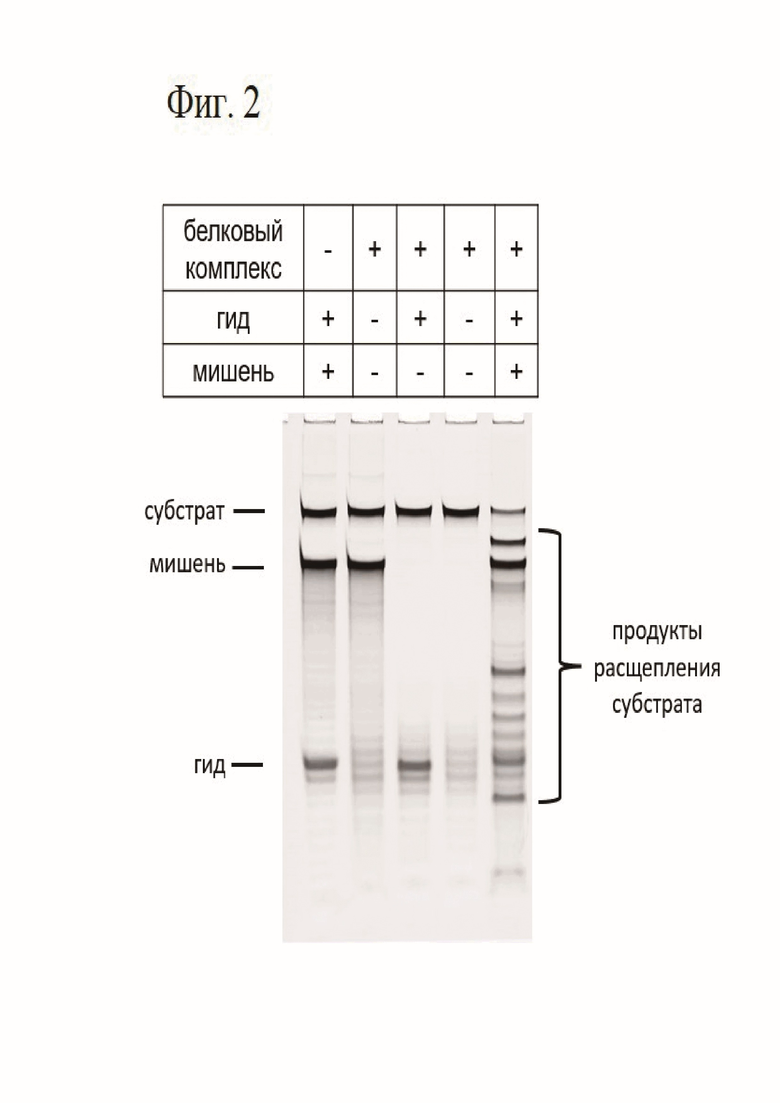
При разработке настоящего изобретения показано, что аргонавт NbaAgo и его белок-партнер DREN при совместной экспрессии формируют стабильный комплекс, что подтверждается электрофоретическим исследованием препаратов (фиг. 1), таким образом их легко использовать совместно. В ходе разработки данного изобретения показано, что в комплекс короткого аргонавта и его белка-партнера, который содержит домен нуклеазы, приобретает нуклеазную активность только при связывании аргонавтом гида и комплементарной гиду мишени (фиг. 2). Таким образом, комплекс белка-аргонавта и его партнера в данном изобретении выступает в качестве сенсора присутствия специфической ДНК.
Данный белок-аргонавт не требует для узнавания целевой ДНК наличия специальной последовательности, как последовательность PAM для систем на основе CRISPR, поэтому его применение возможно для любой целевой детектируемой последовательности на любом ее участке.
В отличие от систем на базе длинного аргонавта археи Pyrococcus furiosus, не нужно для каждой мишени подбирать набор из нескольких гидов и создавать специальный репортер - для тест-системы на основе короткого аргонавта NbaAgo и его белка-партнера DREN достаточно подобрать одну гидовую молекулу РНК и одну репортерную молекулу ДНК. Причем одна и та же репортерная молекула ДНК может быть использована во всех системах на основе этих белков для детекции любых последовательностей, что делает репортерную молекулу ДНК универсальной. Кроме того, нуклеазный центр комплекса NbaAgo и DREN удален от центра связывания гида и узнавания мишени. Это позволяет комплексу, в отличие от аргонавта Pyrococcus furiosus и других длинных аргонавтов, осуществлять расщепление субстрата без обмена уже связанных гида и мишени, т.е. многораундовую реакцию расщепления.
Таким образом, отсутствие требований к специфическим последовательностям, таким как PAM, необходимости создавать для каждой целевой ДНК набор гидов и новый репортер, и способность к многораундному катализу делают тест-системы на основе комплекса белка-аргонавта и его партнера из Novosphingopyxis baekryungensis значительно более универсальными, чем разработанные ранее другие тест-системы.
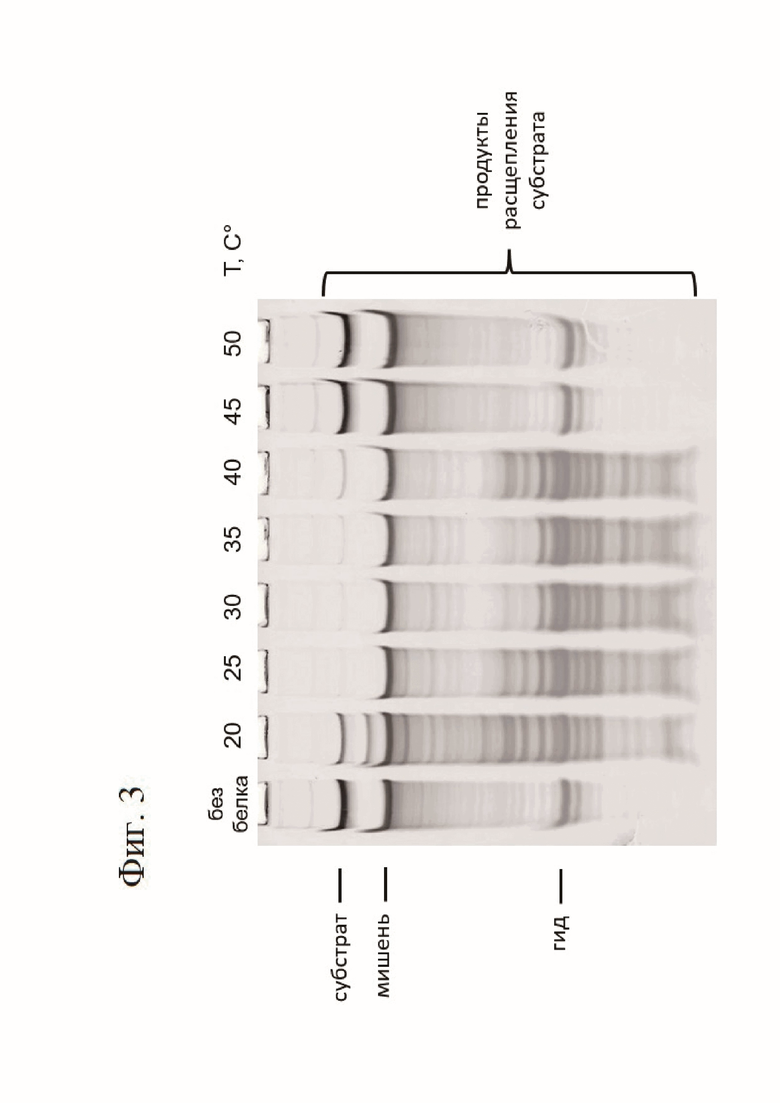
Еще одним преимуществом использования комплекса короткого белка аргонавта и его партнёра является его температурный оптимум активности в физиологическом диапазоне температур - от 20°С до 40°С, что значительно упрощает его применение в полевых условиях (фиг. 3). Такой температурный диапазон позволяет использовать тест-систему на базе комплекса короткого аргонавта без необходимости применения термостатов.
Также использование короткого аргонавта Novophingopyxis baekryungensis в комплексе с DREN позволяет заменить флуоресцентный аналог НАД в качестве репортерной молекулы на двуцепочечную или одноцепочечную ДНК или РНК, содержащую в молекуле флуорофор и гаситель. При расщеплении такой ДНК или РНК появляется детектируемый флуоресцентный сигнал. Преимущества применения ДНК или РНК в качестве репортерной молекулы заключаются в следующем: 1) это значительно дешевле, чем использование дорогого флуоресцентного аналога НАД, 2) можно использовать разнообразные флуоресцентные метки, что не ограничивает каналы детекции сигнала, 3) такая система позволяет применять для детекции сигнала приборы для ПЦР в реальном времени, а также просто ультрафиолетовые лампы, а не только планшетные ридеры, которые представлены не в каждой диагностической лаборатории. Оба эти качества тест-системы на основе комплекса белка-аргонавта и его партнера из Sphingopyxis baekryungensis значительно облегчают ее применение в полевых условиях.
Данная система может быть сопряжена с системой обратной транскрипции и/или с системами изотермальной амплификации для детекции РНК и/или увеличения чувствительности.
Таким образом техническим результатом настоящего изобретения является увеличение разнообразия доступных способов детекции специфических последовательностей ДНК или РНК.
Краткое описание чертежей
Фигура 1. Электрофореграмма препаратов комплекса короткого белка аргонавта и его белка-партнера (NbaAgo-DREN)
Фигура 2. Электрофореграмма продуктов расщепления субстрата комплексом NbaAgo-DREN в присутствии и в отсутствие гида и мишени
Фигура 3. Электрофореграмма продуктов расщепления субстрата комплексом при разных температурах
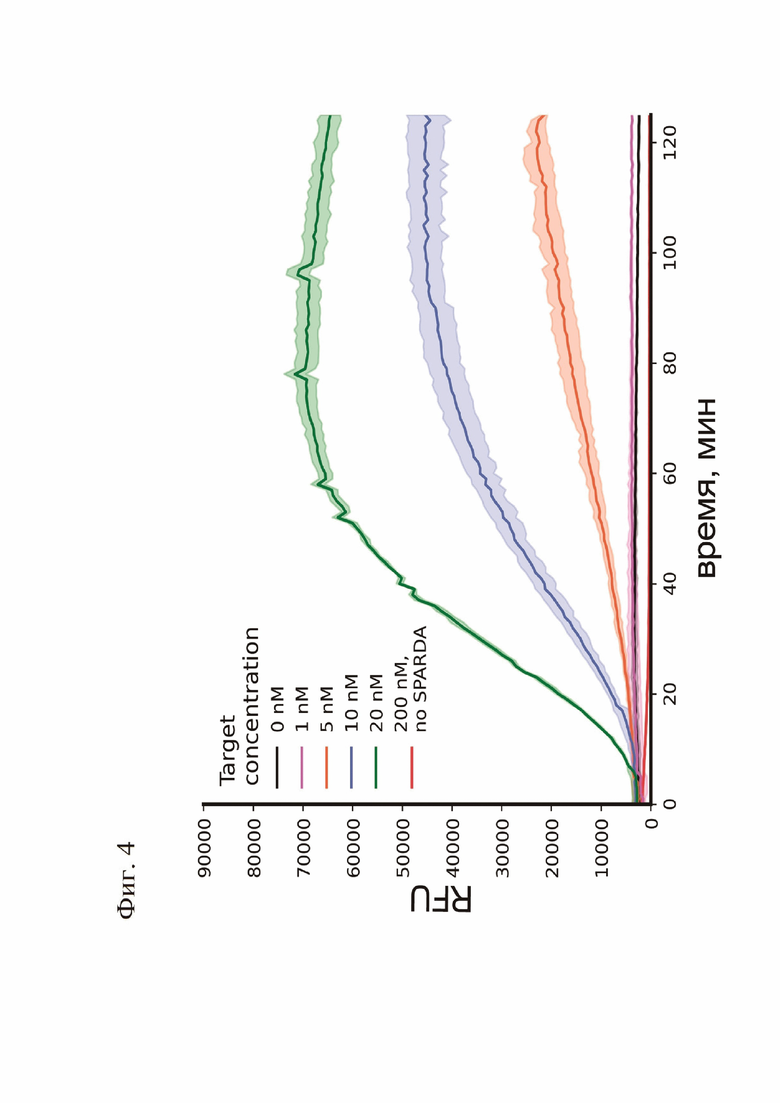
Фигура 4. График накопления флуоресцентного сигнала при использовании в качестве флуоресцентной репортерной молекулы оцДНК (SEQ ID NO: 5), разные концентрации мишени (SEQ ID NO: 4) и планшетного ридера для детекции сигнала
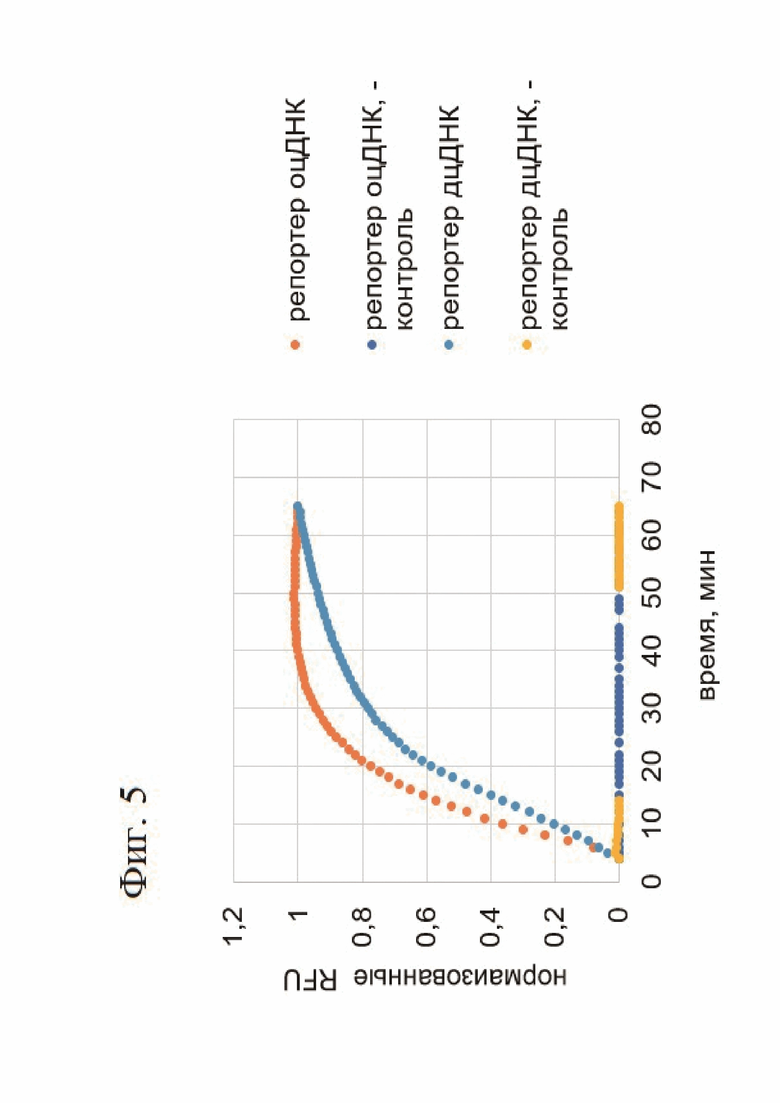
Фигура 5. График накопления флуоресцентного сигнала при использовании в качестве флуоресцентной репортерной молекулы оцДНК и дцДНК (SEQ ID NO: 6-7) и прибора для ПЦР в реальном времени для детекции сигнала
Осуществление изобретения
Если не определено отдельно, технические и научные термины в данной заявке имеют стандартные значения, общепринятые в научной и технической литературе.
Настоящее изобретение и варианты его реализации иллюстрируются приведенными ниже примерами. Доказательствами достижимости технического результата в примере служит: 1) нуклеазная активность препарата комплекса короткого аргонавта, которая наблюдается только в присутствии гида и мишени, но не в контрольных реакциях, где отсутствует мишень или гид (в реакциях in vitro при анализе продуктов расщепления ДНК при помощи гель-электрофореза), а также 2) детекция флуоресцентного сигнала при применении лабораторного прототипа тест-системы с использованием планшетного ридера и 3) детекции флуоресцентного сигнала при применения прибора для проведения ПЦР в реальном времени.
Пример 1. Для проведения всех экспериментов использовали препарат комплекса рекомбинантных белка короткого аргонавта из бактерии Novosphingopyxis baekryungensis (SbAgo) и его белка-партнера DREN, экспрессированного в E.coli. Для экспрессии в E.coli использовали плазмиду pBAD-NbaAgo на базе экспрессионного вектора pBAD, кодирующую оба белка под контролем одного арабинозного промотора и с двумя раздельными сайтами посадки рибосом (последовательность SEQ ID NO: 8). Трансформированные E.coli подращивали в жидкой среде LB до OD600 = 0,5 при 37°С, инкубировали 30 минут во льду, добавляли арабинозу до 0,05% и инкубировали в течении ночи при 16°С. После инкубации клетки осаждали, ресуспендировали в ледяном буферном растворе, содержащем 20 мМ HEPES pH 7.5, 1М KCl, 10 мМ KH2PO4, 5% глицерин, 2 мМ PMSF, 5 мМ β-меркаптоэтанол. Ресуспендированные клетки разрушали обработкой ультразвуком. Лизат осветляли с помощью центрифугирования и наносили на колонку (объем 1 мл) с носителем Ni-сефароза. Колонку промывали тем же буферным раствором (20 мл), но содержащем 0.5М KCl и затем тем же буферным растфором (20 мл), содержащем 0.5М KCl и 20 мМ имидазола. Комплекс NbaAgo-DREN был элюирован тем же буферным растфором, содержащем 300 мМ имидазола. Во фракции, содержащие белок, был добавлен ЭДТА до 5 мМ. Фракции были разведены до концентрации KCl 40 мМ и нанесены на колонку с носителем гепарин-сефароза объемом 5 мл. Колонка была промыта буферным раствором, содержащем 20 мМ HEPES pH 7.5, 40 мМ KCl, 5% глицерин. Комплекс NbaAgo-DREN был элюирован градиентом KCl 40-500 мМ KCl. Фракции, содержащие комплекс, были разведены до концентрации KCl 40 мМ и нанесены на колонку, содержащую носитель monoQ (1 мл). Хроматография была проведена таким же образом, как и предыдущая хроматография на колонке с носителем гепарин-сефароза. Фракции элюции, содержащие комплекс, были сконцентрированы. Был добавлен равный объем глицерина и DTT до 2 мМ. Полученный препарат хранили при -20°С.
Полученный препарат комплекса короткого аргонавта и его партнера (конечная концентрация в реакции 500 нМ, объем реакции 15 мкл) смешивали с гидовой РНК (конечная концентрация в реакции 100 нМ, последовательностью SEQ ID NO: 3) и мишенью (конечная концентрация в реакции 100 нМ, последовательностью SEQ ID NO: 4) в буферном растворе 20 мМ HEPES-KOH pH 7.5, 30 мМ KCl, 5% глицерин, 5 мкг/мл БСА, и инкубировали 15 минут при температуре 30°С. Затем в реакцию, где указано, добавляли ДНК-субстрат длиной 71 нуклеотид (SEQ ID NO: 9) и инкубировали 30 минут при температуре 30°С. Реакцию останавливали добавлением буфера для нанесения и проводили электрофорез в денатурирующих условиях в 19% ПААГ. Результат реакции и контрольных реакций приведен на фигуре 3.
Пример 2. Другим примером достижимости результата является накопление флуоресцентного сигнала, при смешивании белкового комплекса, гида, мишени и репортерной одноцепочечной ДНК (SEQ ID NO: 5, фиг. 4). Был использован препарат белка, полученный также, как в примере 1. Реакции проводили также, как в описанном выше эксперименте, за исключением того, что в качестве субстрата использовали флуоресцентную репортерную оцДНК (SEQ ID NO: 5). Инкубация проводилась в планшетном ридере CLARIOSTAR при температуре 30°С с детекцией флуоресцентного сигнала каждую минуту начиная с добавления субстрата. Результат, представленный на фиг. 4 свидетельствует о том, что чувствительность прототипа тест-системы находится в наномолярном диапазоне.
Пример 3. В третьем примере в аналогичном эксперименте детекция проводилась в приборе для ПЦР в реальном времени BIO-RAD C1000 с использованием в качестве репортерной молекулы дцДНК или оцДНК (SEQ ID NO: 6 и 7 или SEQ ID NO: 5; фиг. 5).
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing originalFreeTextLanguageCode="ru"
dtdVersion="V1_3" fileName="Argonaute.xml" softwareName="WIPO
Sequence" softwareVersion="2.1.2" productionDate="2023-06-15">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText></ApplicationNumberText>
<FilingDate></FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>non</ApplicantFileReference>
<ApplicantName languageCode="ru">Федеральное государственное
бюджетное учреждение науки Институт биологии гена Российской академии
наук </ApplicantName>
<ApplicantNameLatin>Institute of Gene Biology Russian Academy of
Sciences</ApplicantNameLatin>
<InventionTitle languageCode="ru">Тест-система для детекции целевой
ДНК или РНК с использованием короткого белка-аргонавта в комплексе с
нуклеазой и флуоресцентного репортера</InventionTitle>
<SequenceTotalQuantity>9</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>485</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..485</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Novosphingopyxis
baekryungensis</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MTFETRIFDEPELEFGDHHHHQDPRLGLSEAGPLQTFLGDVIKIGVVGN
SKTIEDTRKFIETVSSGVEGKGEKHPNMHPPFPGLGNQSPYRCRFEIEDGATAALTKSKLDKIGKEPDHY
RAVEMAVDEIIGELQAMDDGGSRPDVAIIALPVKLLERVWNAKVDARGTTEKSDSSGSDAPNFRGMLKAK
AMGLSFPIQIVWEDVIDDKVTIPQKVKESSSRKIQDIAGRTWNLMTSLYYKGSGRIPWRRMPLEGEFSAC
YVGISFYREADGQQLFTSAAQMFDERGRGFVLKGRRARTESRGRHPYMAREDAKKIIEDVLAAYKLHHKT
LPARVFILKTSRFKDEEADGIIAALDEAGTELRDLVWVQESYTARILRDGNYPVLRGTFVDLHGKGLLYT
SGSMPYYGTYPGKYDPNPLLLCPHHTSESTVAQLAEEIFSLTKVNWNSTQMNQRLPIPIRAARKVGEVLK
YVGEGEVISADYRKYI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>451</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..451</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q15">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Novosphingopyxis
baekryungensis</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>HHHHHHGSGVTKKITANQIIGEIGENEVRGRFLTLGWQFDGRSRLEAGI
DGIAEVMNEGQPMARMIAVQIKSTKEGKYTSESDTSFTYLLRTQDLAYWRGSNLPVIVVFYRQSDHSFYW
KEVSRDAGPGERRLNIDKVADLFNASTVNKLAALTVPKTGLGYYVPPLGGGEDALINMLPLTLPNEMYIA
STTYEPRKAIAVILNGDGPKRFDWVINGGTFWSFHDPRTSACSEIVDIDQVEAINTKELALHDDIDEQNR
FSHLLRQTLRYQTDSDLGWDKDHKALYFRAIEREVSRNFAYTSSKKKTDANVVSVFKNSKDETRVSFVRH
HAFSPRFELMADQWYLIITPTYYYTTNGYAPHQFAAPLLAGKKRLDKSAALRGQVIMWHRFLTQSDHEDL
FHSEETPEAYLMFGEPPSIHLDVRVPEDGWVKEKVKRIDEAAQGEGLFSDDI</INSDSeq_sequence
>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atactgcacagctgacgata</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>50</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..50</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q5">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tccattgtgtgttgtatcgtcagctgtgcagtattgtagttgttgttct
t</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>32</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..32</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q7">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>3'UTR</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q18">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>BHQ2</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>5'UTR</INSDFeature_key>
<INSDFeature_location>>32</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q19">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>ROX</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtcacagagatactacgtgtgcgactgctcag</INSDSeq_sequenc
e>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>32</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..32</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q9">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>3'UTR</INSDFeature_key>
<INSDFeature_location>>32</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q17">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>ROX</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtcacagagatactacgtgtgcgactgctcag</INSDSeq_sequenc
e>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>32</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..32</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>3'UTR</INSDFeature_key>
<INSDFeature_location>>32</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q16">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>BHQ2</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctgagcagtcgcacatgtagtatctctgtgac</INSDSeq_sequenc
e>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>6833</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..6833</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q3">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aagaaaccaattgtccatattgcatcagacattgccgtcactgcgtctt
ttactggctcttctcgctaaccaaaccggtaaccccgcttattaaaagcattctgtaacaaagcgggacc
aaagccatgacaaaaacgcgtaacaaaagtgtctataatcacggcagaaaagtccacattgattatttgc
acggcgtcacactttgctatgccatagcatttttatccataagattagcggatcctacctgacgcttttt
atcgcaactctctactgtttctccatacccgttttttgggctaacaggaggtatcaggatgcatcaccac
catcatcacggctctggcgtgaccaaaaaaatcactgccaatcagatcattggggagatcggcgaaaatg
aggtccgtggacggtttctgacactaggctggcagttcgatggacggtccagattggaagcgggcattga
tgggatcgcggaagtcatgaacgagggccaaccaatggcccgcatgatcgcagtgcaaataaagtccacc
aaggaggggaaatatacctccgaatctgacacgagcttcacttaccttttgcgcacccaagaccttgcct
attggagagggtcaaaccttccagtcatcgtcgttttttacagacagtcggaccacagtttctactggaa
agaagtgtcgcgggatgctggtcccggcgagcggcgactgaatatcgacaaggtcgcggacctctttaac
gctagcaccgtgaacaaattggccgcattgactgtcccaaaaaccggcctgggatattacgtgcctccgc
tcggcggtggtgaagatgcattgatcaatatgctcccacttacgcttccaaatgaaatgtatatcgcttc
gacgacatacgagccacggaaggctatagcggtcattcttaatggagatggcccaaagcgcttcgactgg
gtgatcaatggcggaacgttctggtctttccatgaccctagaacttcagcatgctcagagattgtggata
tcgatcaagtggaagctatcaacactaaagagttggcacttcatgacgatatcgatgagcaaaatagatt
tagccacctccttcgccaaacgctccggtatcaaactgattctgacctcggttgggataaggatcacaaa
gccctttattttcgcgcaattgaaagagaagtttcccgtaattttgcttacacatcatctaaaaagaaaa
ccgacgctaacgttgttagcgtatttaaaaactcaaaagatgagaccagggtttcctttgtgcgccacca
tgccttcagcccccggttcgaactcatggcagatcagtggtatctaatcataactcctacgtattattat
acgacgaatggatatgctccgcatcaatttgctgccccgcttttggcagggaaaaaacggctcgataaaa
gcgcagctctgagggggcaggtcattatgtggcatcgctttctcactcaaagcgatcacgaagatttatt
ccatagtgaagaaacgccagaggcgtatttgatgtttggcgaaccgccttcaattcatttggatgtaagg
gttcctgaagatggctgggtgaaggaaaaggtcaaaaggatcgacgaggcggctcagggtgaggggttgt
tttccgatgacatttaataacggatccgaattcgagctccgtcgacaagcaaggagatatacatatgaca
tttgaaacgcgcattttcgatgagccagaactggaatttggcgatcatcatcatcaccaagatccacgcc
tgggcttatcagaggcgggaccgcttcaaaccttcctaggggatgtcataaaaataggcgttgttggaaa
ctccaagacaatcgaggacactcgaaaattcatcgagaccgtcagtagcggtgttgagggtaagggcgaa
aaacaccccaacatgcacccgccttttccgggacttggcaatcaaagtccatatcgatgccgatttgaga
tcgaagacggtgcaacggctgcgttgaccaagagcaaactcgataagatcggaaaggagcccgaccacta
ccgcgctgtggaaatggcagtcgacgaaatcattggcgaactgcaggctatggatgatggcggaagccgt
cctgacgttgcaatcatagcactgccagtaaagcttctggagcgcgtatggaacgcaaaagtcgatgctc
gtgggacaaccgagaagtcggacagcagtggctctgacgctcctaattttcgcggaatgctaaaagcgaa
agcaatggggctaagttttccaattcaaattgtttgggaagatgtgatcgacgacaaggttacgatcccg
cagaaggtcaaagaaagtagctctcgcaaaatccaggatattgccggtcgcacatggaacctaatgacca
gcctctattacaaagggagcggacgtattccttggcgccgtatgccattggagggcgaattttctgcctg
ctacgtcggaataagcttctaccgtgaagcagacggccaacaactttttaccagtgcggcccagatgttc
gacgagcgcggccgaggttttgtattgaagggaaggcgcgcacggaccgagagtcgcggcaggcatccat
acatggctcgtgaagatgccaaaaaaattatagaggatgttctggctgcctataagcttcatcataagac
cctgccagcccgcgtattcatcctgaagacctcacgttttaaggacgaggaagcggacggaataattgct
gcattggatgaggctggcaccgaattacgcgatctggtttgggttcaagagtcctatactgcacggatcc
tacgcgatggaaattatcccgtcttgcgcggcacgtttgttgatttgcatggtaagggattgctctacac
tagcggaagcatgccttactacggcacctaccctggcaaatacgatcctaatccgctcttgctgtgcccg
catcataccagtgaaagcacggtcgcccagctggcagaagaaattttctcactgacaaaagtgaattgga
attccacacaaatgaaccaacgtcttccgatcccaatcagggctgcgcgaaaggtgggtgaggtcctaaa
atatgttggtgagggcgaagtcatcagcgcggattatcggaaatatatttgatagatgggaattcgaagc
ttggctgttttggcggatgagagaagattttcagcctgatacagattaaatcagaacgcagaagcggtct
gataaaacagaatttgcctggcggcagtagcgcggtggtcccacctgaccccatgccgaactcagaagtg
aaacgccgtagcgccgatggtagtgtggggtctccccatgcgagagtagggaactgccaggcatcaaata
aaacgaaaggctcagtcgaaagactgggcctttcgttttatctgttgtttgtcggtgaacgctctcctga
gtaggacaaatccgccgggagcggatttgaacgttgcgaagcaacggcccggagggtggcgggcaggacg
cccgccataaactgccaggcatcaaattaagcagaaggccatcctgacggatggcctttttgcgtttcta
caaactcttttgtttatttttctaaatacattcaaatatgtatccgctcatgagacaataaccctgataa
atgcttcaataatattgaaaaaggaagagtatgagtattcaacatttccgtgtcgcccttattccctttt
ttgcggcattttgccttcctgtttttgctcacccagaaacgctggtgaaagtaaaagatgctgaagatca
gttgggtgcacgagtgggttacatcgaactggatctcaacagcggtaagatccttgagagttttcgcccc
gaagaacgttttccaatgatgagcacttttaaagttctgctatgtggcgcggtattatcccgtgttgacg
ccgggcaagagcaactcggtcgccgcatacactattctcagaatgacttggttgagtactcaccagtcac
agaaaagcatcttacggatggcatgacagtaagagaattatgcagtgctgccataaccatgagtgataac
actgcggccaacttacttctgacaacgatcggaggaccgaaggagctaaccgcttttttgcacaacatgg
gggatcatgtaactcgccttgatcgttgggaaccggagctgaatgaagccataccaaacgacgagcgtga
caccacgatgcctgtagcaatggcaacaacgttgcgcaaactattaactggcgaactacttactctagct
tcccggcaacaattaatagactggatggaggcggataaagttgcaggaccacttctgcgctcggcccttc
cggctggctggtttattgctgataaatctggagccggtgagcgtgggtctcgcggtatcattgcagcact
ggggccagatggtaagccctcccgtatcgtagttatctacacgacggggagtcaggcaactatggatgaa
cgaaatagacagatcgctgagataggtgcctcactgattaagcattggtaactgtcagaccaagtttact
catatatactttagattgatttaaaacttcatttttaatttaaaaggatctaggtgaagatcctttttga
taatctcatgaccaaaatcccttaacgtgagttttcgttccactgagcgtcagaccccgtagaaaagatc
aaaggatcttcttgagatcctttttttctgcgcgtaatctgctgcttgcaaacaaaaaaaccaccgctac
cagcggtggtttgtttgccggatcaagagctaccaactctttttccgaaggtaactggcttcagcagagc
gcagataccaaatactgtccttctagtgtagccgtagttaggccaccacttcaagaactctgtagcaccg
cctacatacctcgctctgctaatcctgttaccagtggctgctgccagtggcgataagtcgtgtcttaccg
ggttggactcaagacgatagttaccggataaggcgcagcggtcgggctgaacggggggttcgtgcacaca
gcccagcttggagcgaacgacctacaccgaactgagatacctacagcgtgagctatgagaaagcgccacg
cttcccgaagggagaaaggcggacaggtatccggtaagcggcagggtcggaacaggagagcgcacgaggg
agcttccagggggaaacgcctggtatctttatagtcctgtcgggtttcgccacctctgacttgagcgtcg
atttttgtgatgctcgtcaggggggcggagcctatggaaaaacgccagcaacgcggcctttttacggttc
ctggccttttgctggccttttgctcacatgttctttcctgcgttatcccctgattctgtggataaccgta
ttaccgcctttgagtgagctgataccgctcgccgcagccgaacgaccgagcgcagcgagtcagtgagcga
ggaagcggaagagcgcctgatgcggtattttctccttacgcatctgtgcggtatttcacaccgcatatgg
tgcactctcagtacaatctgctctgatgccgcatagttaagccagtatacactccgctatcgctacgtga
ctgggtcatggctgcgccccgacacccgccaacacccgctgacgcgccctgacgggcttgtctgctcccg
gcatccgcttacagacaagctgtgaccgtctccgggagctgcatgtgtcagaggttttcaccgtcatcac
cgaaacgcgcgaggcagcagatcaattcgcgcgcgaaggcgaagcggcatgcataatgtgcctgtcaaat
ggacgaagcagggattctgcaaaccctatgctactccgtcaagccgtcaattgtctgattcgttaccaat
tatgacaacttgacggctacatcattcactttttcttcacaaccggcacggaactcgctcgggctggccc
cggtgcattttttaaatacccgcgagaaatagagttgatcgtcaaaaccaacattgcgaccgacggtggc
gataggcatccgggtggtgctcaaaagcagcttcgcctggctgatacgttggtcctcgcgccagcttaag
acgctaatccctaactgctggcggaaaagatgtgacagacgcgacggcgacaagcaaacatgctgtgcga
cgctggcgatatcaaaattgctgtctgccaggtgatcgctgatgtactgacaagcctcgcgtacccgatt
atccatcggtggatggagcgactcgttaatcgcttccatgcgccgcagtaacaattgctcaagcagattt
atcgccagcagctccgaatagcgcccttccccttgcccggcgttaatgatttgcccaaacaggtcgctga
aatgcggctggtgcgcttcatccgggcgaaagaaccccgtattggcaaatattgacggccagttaagcca
ttcatgccagtaggcgcgcggacgaaagtaaacccactggtgataccattcgcgagcctccggatgacga
ccgtagtgatgaatctctcctggcgggaacagcaaaatatcacccggtcggcaaacaaattctcgtccct
gatttttcaccaccccctgaccgcgaatggtgagattgagaatataacctttcattcccagcggtcggtc
gataaaaaaatcgagataaccgttggcctcaatcggcgttaaacccgccaccagatgggcattaaacgag
tatcccggcagcaggggatcattttgcgcttcagccatacttttcatactcccgccattcagag</INSD
Seq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>71</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..71</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agctaggatcgctccgatggttcgagtgatagatcgcttgttcacacag
gaaacagcgcattgccctagtg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Средство обнаружения нуклеиновых кислот на основе ScCas12a белка из бактерии Sedimentisphaera cyanobacteriorum | 2023 |
|
RU2820345C1 |
| Система CRISPR-Cas12 для выявления РНК вируса гепатита С генотипов 1b и 3a в ультранизких концентрациях | 2022 |
|
RU2800421C1 |
| Способ получения препарата рибонуклеопротеинового комплекса CRISPR-Cas и препарат для выявления РНК вируса гепатита С генотипов 1b и 3a в ультранизких концентрациях | 2022 |
|
RU2799430C1 |
| Набор CRISPR-Cas12 для выявления гена антибиотикоустойчивости bla-NDM-1 в ультранизких концентрациях (варианты) | 2024 |
|
RU2839482C1 |
| Способ обнаружения РНК вируса гепатита С генотипов 1b и 3a в ультранизких концентрациях и специфические олигонуклеотиды для использования в способе | 2022 |
|
RU2800420C1 |
| Способ обнаружения РНК вируса иммунодефицита человека (ВИЧ-1) в ультранизких концентрациях и специфические олигонуклеотиды для использования в способе | 2022 |
|
RU2802079C1 |
| Система CRISPR-Cas12 для выявления гена антибиотикоустойчивости bla-NDM-1 в ультранизких концентрациях | 2024 |
|
RU2839761C1 |
| Способ получения препарата рибонуклеопротеинового комплекса CRISPR-Cas12 и препарат для выявления РНК вируса иммунодефицита человека (ВИЧ-1) в ультранизких концентрациях | 2022 |
|
RU2802781C1 |
| Способ получения препарата рибонуклеопротеинового комплекса CRISPR-Cas и препарат для выявления гена антибиотикоустойчивости bla-NDM-1 в ультранизких концентрациях | 2024 |
|
RU2839484C1 |
| Способ обнаружения гена антибиотикоустойчивости bla-NDM-1 в ультранизких концентрациях и специфические олигонуклеотиды для использования в способе | 2024 |
|
RU2841369C1 |
Изобретение относится к области биотехнологии. Описана группа изобретений, включающая тест-систему на основе комплекса для детекции целевых молекул нуклеиновых кислот, применение комплекса для детекции целевых ДНК или РНК и способ определения наличия в образце молекулы нуклеиновой кислоты-мишени. В одном из вариантов реализации тест-система содержит комплекс, основанный на коротком белке-аргонавте из бактерии Novosphingopyxis baekryungensis (NbaAgo), имеющем аминокислотную последовательность SEQ ID NO: 1, его белке-партнере DREN из бактерии Novosphingopyxis baekryungensis, имеющем аминокислотную последовательность SEQ ID NO: 2, а также направляющей РНК, молекулы нуклеиновой кислоты-мишени и универсального флуоресцентного репортера. Изобретение расширяет арсенал средств для детекции целевых молекул нуклеиновых кислот. 3 н. и 7 з.п. ф-лы, 5 ил., 3 пр.
1. Тест-система для детекции целевых молекул нуклеиновых кислот на основе комплекса, состоящего из:
1) короткого белка-аргонавта из бактерии Novosphingopyxis baekryungensis (NbaAgo), имеющего аминокислотную последовательность SEQ ID NO: 1;
2) его белка-партнера DREN из бактерии Novosphingopyxis baekryungensis, имеющего аминокислотную последовательность SEQ ID NO: 2;
а также направляющей РНК, молекулы нуклеиновой кислоты-мишени и универсального флуоресцентного репортера – флуоресцентной репортерной нуклеиновой кислоты, причем флуоресцентная репортерная нуклеиновая кислота имеет флуоресцентную группу и гасящую группу.
2. Тест-система по п.1, в которой в качестве направляющей РНК используется последовательность SEQ ID NO: 3, в качестве мишени – последовательность SEQ ID NO: 4.
3. Тест-система по п.1, в которой в качестве флуоресцентной репортерной нуклеиновой кислоты используется оцДНК.
4. Тест-система по п.1, в которой в качестве флуоресцентной репортерной нуклеиновой кислоты используется дцДНК.
5. Применение для детекции целевых ДНК или РНК комплекса, состоящего из:
1) короткого белка-аргонавта из бактерии Novosphingopyxis baekryungensis (NbaAgo), имеющего аминокислотную последовательность SEQ ID NO: 1;
2) его белка-партнера DREN из бактерии Novosphingopyxis baekryungensis, имеющего аминокислотную последовательность SEQ ID NO: 2;
а также направляющей РНК, молекулы нуклеиновой кислоты-мишени и универсального флуоресцентного репортера – флуоресцентной репортерной нуклеиновой кислоты, причем флуоресцентная репортерная нуклеиновая кислота имеет флуоресцентную группу и гасящую группу.
6. Применение по п.5, где в качестве направляющей РНК используется последовательность SEQ ID NO: 3, в качестве мишени – последовательность SEQ ID NO: 4.
7. Применение по п.5, где в качестве флуоресцентной репортерной нуклеиновой кислоты используется оцДНК.
8. Применение по п.5, где в качестве флуоресцентной репортерной нуклеиновой кислоты используется дцДНК.
9. Применение по п.5 при температуре от 20°С до 40°С.
10. Способ определения наличия в образце молекулы нуклеиновой кислоты-мишени, включающий следующие этапы:
(a) предоставление тест-системы для детекции по п.1 для обнаружения молекул нуклеиновой кислоты-мишени;
(b) взаимодействие тест-системы для детекции по п.1 с подлежащим обнаружению образцом при температуре от 20°С до 40°С, в результате чего образуется реакционный раствор;
(c) детекция флуоресценции в реакционном растворе;
при этом, если в реакционном растворе определяется значение сигнала флуоресценции, это означает, что целевая молекула нуклеиновой кислоты существует в образце; а если значение сигнала флуоресценции не определяется в реакционном растворе, это означает, что мишень не существует в молекулах нуклеиновой кислоты образца.
| БД "GenBank" последовательность под номером WP_022673743.1 | |||
| БД "GenBank" последовательность под номером WP_033317603.1 | |||
| WO 2016028843 A2, 25.02.2016 | |||
| Prostova M | |||
| et al | |||
| DNA-targeting short Argonaute triggers effector nuclease to protect bacteria from invaders //bioRxiv, 08.06.2023, p | |||
| 544223 | |||
| Тест-система для выявления специфических нуклеотидных последовательностей, характерных для выявляемых микроорганизмов или вирусов, способ применения тест-системы (варианты) | 2021 |
|
RU2769572C1 |
| Тест-система для выявления SARS-CoV-2, Influenza virus A, Influenza virus B методом одношаговой полимеразной цепной реакции с обратной транскрипцией | 2021 |
|
RU2761481C1 |
Авторы
Даты
2024-06-28—Публикация
2023-06-19—Подача