Область техники
Изобретение относится к области генной инженерии и биотехнологии. А именно - к новым ферментам Cas нуклеазам систем CRISPR-Cas, проявляющим коллатеральную активность в отношении неспецифических молекул нуклеиновых кислот в результате связывания с целевыми молекулами. В качестве неспецифических молекул могут выступать специализированные детектирующие конструкции. Данная технология может применяться в будущем для детекции геномов различных организмов, в том числе для определения бактерий и вирусов при диагностике заболеваний человека.
Работа выполнена при финансовой поддержке Министерства Науки и Высшего образования Российской Федерации в рамках Соглашения № 075-15-2019-1661 от 31.10.2019.
Уровень техники
На сегодняшний день применение технологий молекулярной биологии является востребованным при создании диагностических систем для выявления и генотипирования возбудителей инфекций. В основе систем, наиболее часто применяемых в клинической практике, как правило, лежат методы полимеразной цепной реакции (ПЦР), секвенирования, иммунохроматографического анализа (ИХА). Данные методы обладают значительными преимуществами в сравнении с классическими подходами к идентификации возбудителей, например, с культивированием при использовании селективных диагностических питательных сред. Однако и они не лишены недостатков. Описанные методы требуют значительного количества времени и ручного труда, либо теряют в точности результатов. Кроме того, все эти подходы достаточно трудоемко адаптировать под детекцию нескольких возбудителей в одном образце, используя один универсальный тест.
Одним из перспективных направлений, позволяющих решить данные проблемы, является использование Cas нуклеаз систем CRISPR-Cas.
По своей природе CRISPR-Cas являются иммунными системами прокариот, способными высоко специфично вносить разрывы в генетический материал вирусов (Mojica F. J. M., Díez-Villaseñor C., García-Martínez J. & Soria E. Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements // Journal of molecular evolution. - 2005. - Т. 60. - №. 2. - С. 174-182). Аббревиатура CRISPR-Cas расшифровывается как «Clustered Regularly Interspaced Short Palindromic Repeats and CRISPR associated genes» (Jansen R., Embden J. D., Gaastra W. & Schouls L. M. Identification of genes that are associated with DNA repeats in prokaryotes // Molecular microbiology. - 2002. - Т. 43. - №. 6. - С. 1565-1575), что в переводе с английского обозначает «короткие палиндромные повторы, регулярно расположенные группами, и ассоциированные с ними гены». Все CRISPR-Cas системы состоят из CRISPR кассет и генов, кодирующих различные Cas белки. CRISPR кассеты состоят из последовательностей спейсеров, каждый из которых имеет уникальную нуклеотидную последовательность, и повторяющихся палиндромных повторов (Jansen R., Embden J. D., Gaastra W. & Schouls L. M. Identification of genes that are associated with DNA repeats in prokaryotes // Molecular microbiology. - 2002. - Т. 43. - №. 6. - С. 1565-1575). В результате транскрипции CRISPR кассет и их последующего процессинга образуются направляющие крРНК, которые вместе с Cas белками формируют эффекторный комплекс (Brouns S. J., Jore M. M., Lundgren M., Westra E. R., Slijkhuis R. J., Snijders A. P., Dickman M. J., Makarova K. S., Koonin E. V. & van der Oost J. Small CRISPR RNAs guide antiviral defense in prokaryotes // Science. - 2008. - Т. 321. - №. 5891. - С. 960-964). За счет комплементарного спаривания крРНК с целевым участком ДНК, именуемым протоспейсером, Cas-нуклеаза узнает ДНК-мишень и высоко специфично вносит в нее разрыв.
Таким образом, CRISPR-Cas нуклеазы узнают и расщепляют конкретные мишени за счет использования направляющих РНК, комплементарных целевой последовательности. Это делает CRISPR-Cas системы удобными и эффективными инструментами специфичного расщепления различных молекул нуклеиновых кислот.
В зависимости от типа эффекторов все CRISPR-Cas системы разделяют на два класса. И далее эти классы подразделяются на шесть различных типов (от I до VI). CRISPR-Cas системы класса 2 представлены одиночным белком-эффектором (Makarova K. S., Wolf Y. I., Iranzo J., Shmakov S. A., Alkhnbashi O. S., Brouns S. J. J., Charpentier E., Cheng D., Haft D. H., Horvath P., Moineau S., Mojica F. J. M., Scott D., Shah S. A., Siksnys V., Terns M. P., Venclovas Č., White M. F., Yakunin A. F., Yan W., Zhang F., Garrett R. A., Backofen R., van der Oost J., Barrangou R. & Koonin E. V. Evolutionary classification of CRISPR-Cas systems: a burst of class 2 and derived variants // Nature Reviews Microbiology. - 2020. - Т. 18. - №2. - С. 67-83).
CRISPR системы с эффекторным белком Cas12a (ранее обозначаемым как cpf1) являются представителями класса 2, типа V, подтипа A. Системы данного типа отличаются простотой состава и механизма работы. В отличие от CRISPR-Cas систем II типа с эффекторной нуклеазой Cas9 для их функционирования необходимо формирование эффекторного комплекса, помимо самой нуклеазы включающего только одну направляющую РНК - крРНК (crRNA). Трансактивирующая крРНК (tracrRNA, тракрРНК), комплементарно спаривающаяся с участком крРНК, при этом отсутствует (Zetsche B., Gootenberg J. S., Abudayyeh O. O., Slaymaker I. M., Makarova K. S., Essletzbichler P., Volz S. E., Joung J., van der Oost J., Regev A., Koonin E. V. & Zhang F. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system // Cell. - 2015. - Т. 163. - № 3. - С. 759-771).
Эффекторному рибонуклеиновому комплексу, состоящему из Cas12a и крРНК, для распознавания и последующего гидролиза ДНК помимо комплементарного соответствия спейсера крРНК и протоспейсера необходимо присутствие PAM (от англ. «PAM» - Protospacer Adjusted Motif) на ДНК мишени (Shah S. A., Erdmann S., Mojica F. J. M. & Garrett R. A. Protospacer recognition motifs // RNA Biology. - 2013. - Т. 10. - № 5. - С. 891-899). PAM представляет собой строго определенную последовательность из нескольких нуклеотидов, расположенных в случае CRISPR-Cas систем V-A типа вплотную к 5'-концу протоспейсера на нетаргетной цепи. При отсутствии PAM гидролиза связей в ДНК с образованием двунитевого разрыва не происходит.
Для нуклеаз, относящихся к Cas12a, в отличие от Cas9 белков дополнительно также была показана способность неспецифически гидролизовать одноцепочечную ДНК после распознавания своей целевой ДНК-мишени. Данное свойство получило название «коллатеральная активность» (Chen J. S., Ma E., Harrington L. B., Da Costa M., Tian X., Palefsky J. M. & Doudna J. A. CRISPR-Cas12a target binding unleashes indiscriminate single-stranded DNase activity // Science. - 2018. - Т. 360. - № 6387. - С. 436-439).
Именно это свойство и было предложено для диагностики определенных нуклеиновых кислот в образце (например, связанных с конкретными патогенами). Детекция в данном случае происходит за счет неспецифически расщепляемых одноцепочных молекул ДНК (зондов), в ответ на узнавание нуклеазой Cas12a целевой мишени в геноме возбудителя заболевания.
На сегодняшний день описано порядка 40 различных нуклеаз V-A типа (ArCas12a, AsCas12a, BsCas12a, CeCas12a, FnCas12a, HkCas12a, LbCas12a, MbCas12a, Mb2Cas12a, Mb3Cas12a, PiCas12a, PrCas12a, PxCas12a, TsCas12a и т. д.).
Однако до сих пор сохраняется потребность в характеризации новых эффекторах данной группы. Это может позволить более точно подбирать инструмент под конкретные диагностические системы с учетом различных свойств данных нуклеаз (эффективность расщепления, специфичность, чувствительность к заменам в последовательностях мишени и PAM).
Сущность изобретения
Сущностью настоящего изобретения является создание новых инструментов обнаружения нуклеиновых кислот на основе системы CRISPR-Cas V-A типа. Поиск новых ферментов Cas12a позволит существенно расширить арсенал имеющихся средств детекции. Для решения этой задачи авторами было показано наличие коллатеральной активности Cas12a эффектора ранее охарактеризованной системы из бактерии Sedimentisphaera cyanobacteriorum L21-RPul-D3.
Указанная задача решается путем применения белка, содержащего аминокислотную последовательность SEQ ID NO: 1, или содержащего аминокислотную последовательность, которая по меньшей мере на 95% идентична аминокислотной последовательности SEQ ID NO: 1 и имеет отличия по сравнению с SEQ ID NO: 1 только в неконсервативных аминокислотных остатках, для неспецифического расщепления одноцепочечных молекул ДНК после связывания с целевой мишенью на молекуле ДНК, расположенной непосредственно после нуклеотидной последовательности 5'-(T/C)TTN-3'.
Техническим результатом настоящего изобретения является создание нового инструмента определения специфических последовательностей нуклеиновых кислот на основе коллатеральной активности нуклеазы ScCas12a.
Способ детектирования определенных нуклеиновых кислот различных организмов, включает использование: а) белка, содержащего аминокислотную последовательность SEQ ID NO: 1, б) направляющей РНК, содержащей последовательность, образующую дуплекс с нуклеотидной последовательностью целевой молекулы ДНК, непосредственно примыкающей к нуклеотидной последовательности 5'-(T/C)TTN-3', в) зонды, включающие короткие модифицированные олигонуклеотиды с присоединенным флуорофором FAM и гасителем BHQ1, разделенные нуклеотидной последовательностью: FAM-TTATTATT-BHQ1; при этом взаимодействие указанного белка с направляющей РНК и нуклеотидной последовательностью 5'-(T/C)TTN-3' приводит к образованию двунитевого разрыва в последовательности целевой ДНК, непосредственно примыкающей к последовательности 5'-(T/C)TTN-3', и неспецифическому расщеплению зондов.
Применение предложенной технологии делает возможным создание современных диагностических систем, обладающих высокой чувствительностью и скоростью, сниженной стоимостью, возможностью проведения диагностики в отсутствии специализированного высокотехнологичного оборудования.
Краткое описание рисунков
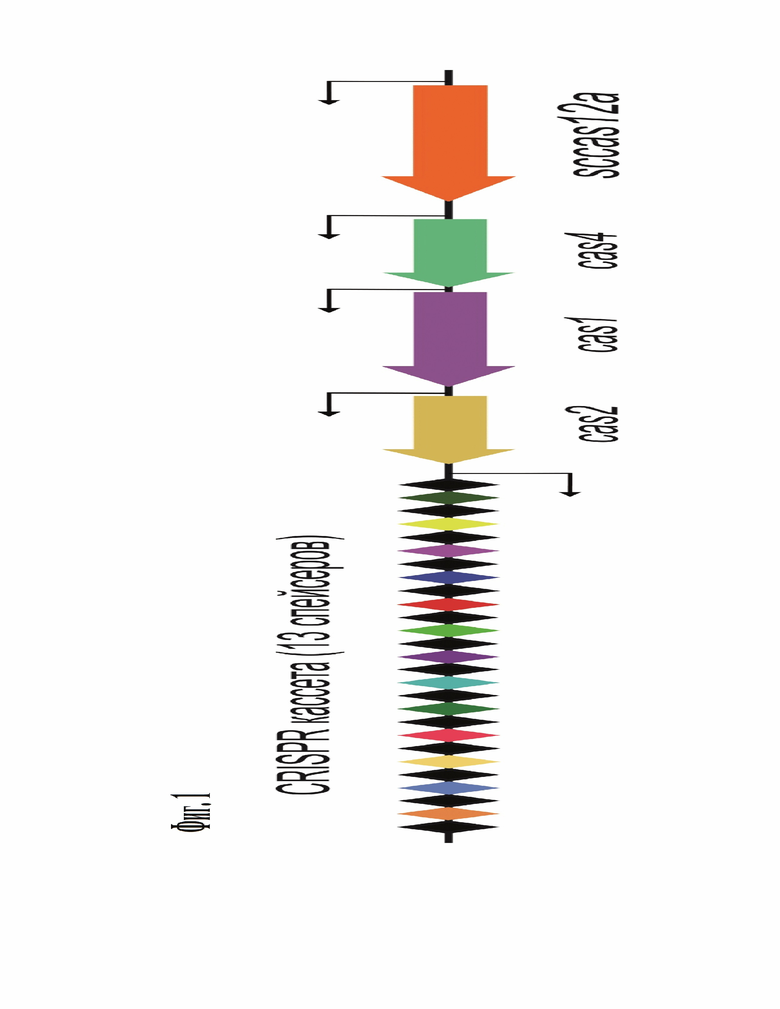
Фиг. 1. Схема устройства локуса CRISPR-Cas системы типа V-A из бактерии Sedimentisphaera cyanobacteriorum.
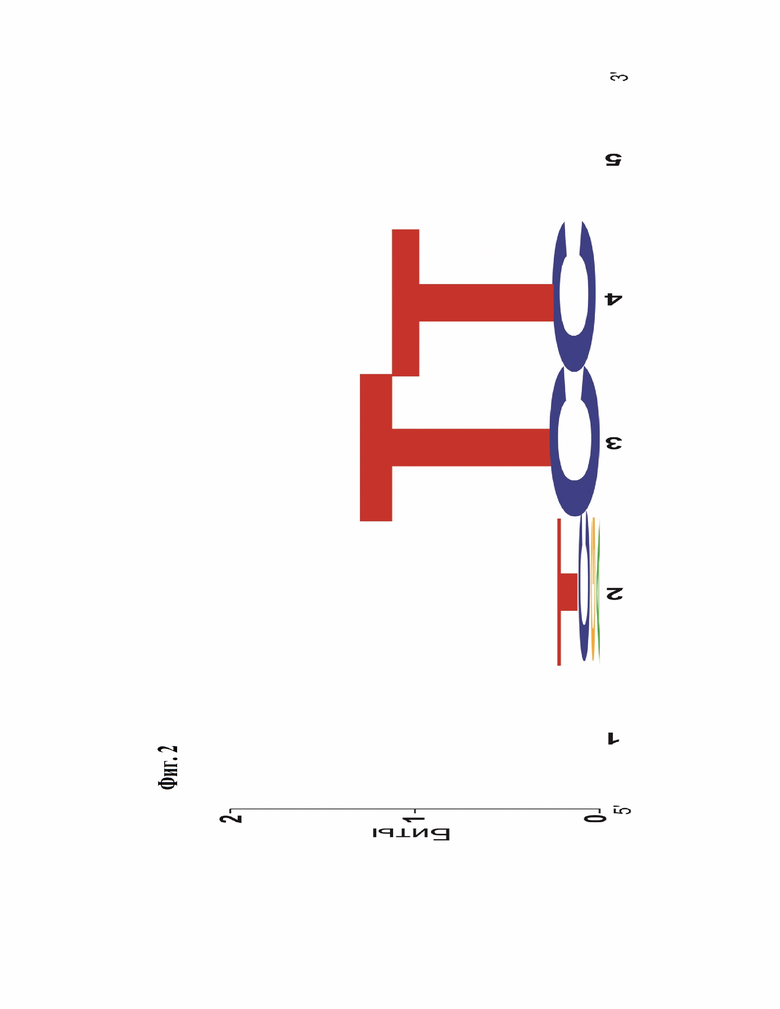
Фиг. 2. PAM Logo для нуклеазы ScCas12a, полученное в результате in vitro деплеционного теста 5N PAM библиотек.
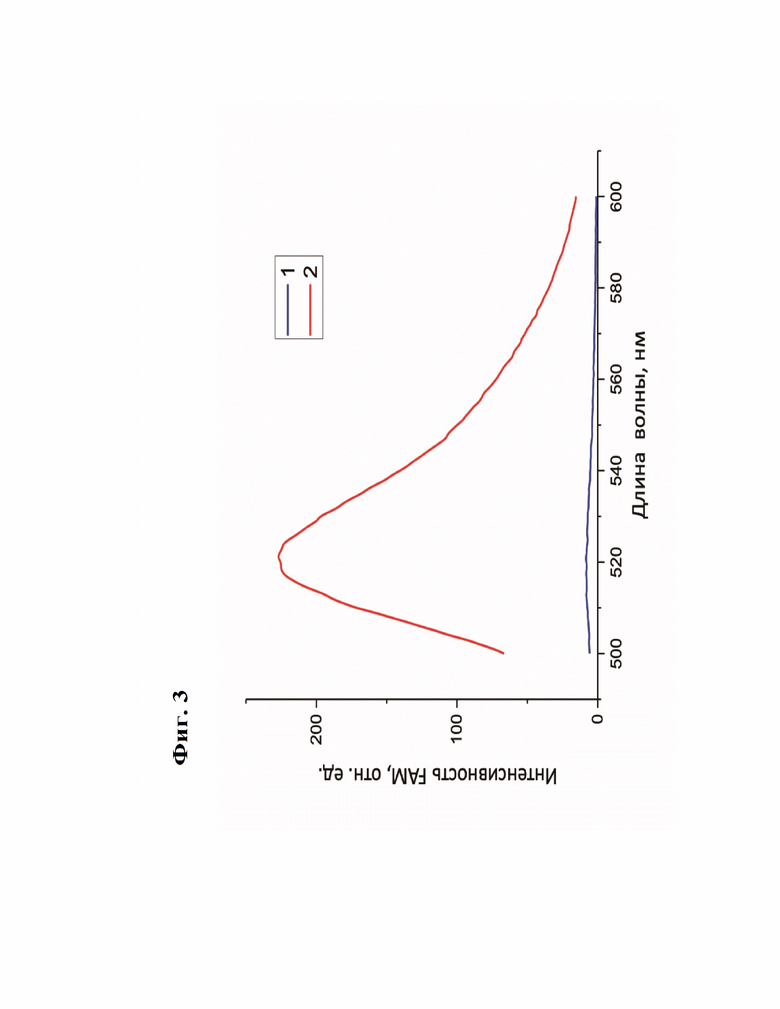
Фиг. 3. Эмиссионные спектры зондов FAM-TTATTATT-BHQ1 при возбуждении излучением 470 нм после проведения реакции. Кривая 1 - контрольные условия в отсутствии белка ScCas12a. Кривая 2 - в присутствии ScCas12a; зонды FAM-TTATTATT-BHQ1 подверглись полному расщеплению.
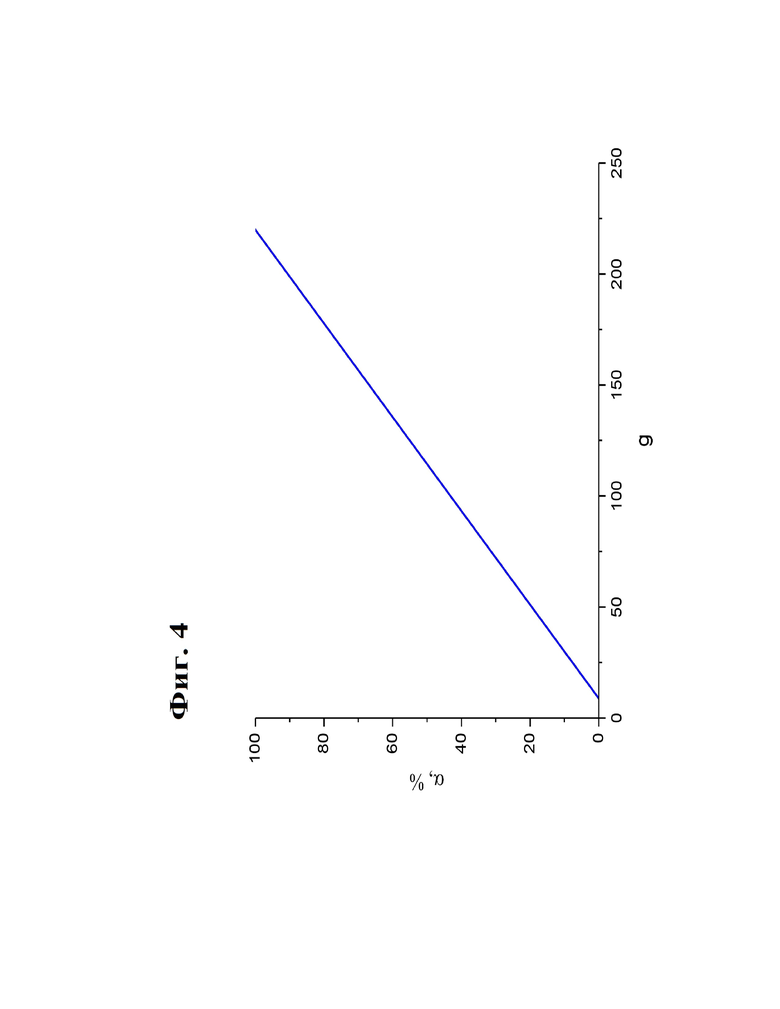
Фиг. 4. Зависимость относительного количества нуклеотидов, подвергшихся расщеплению, от интенсивности свечения FAM.
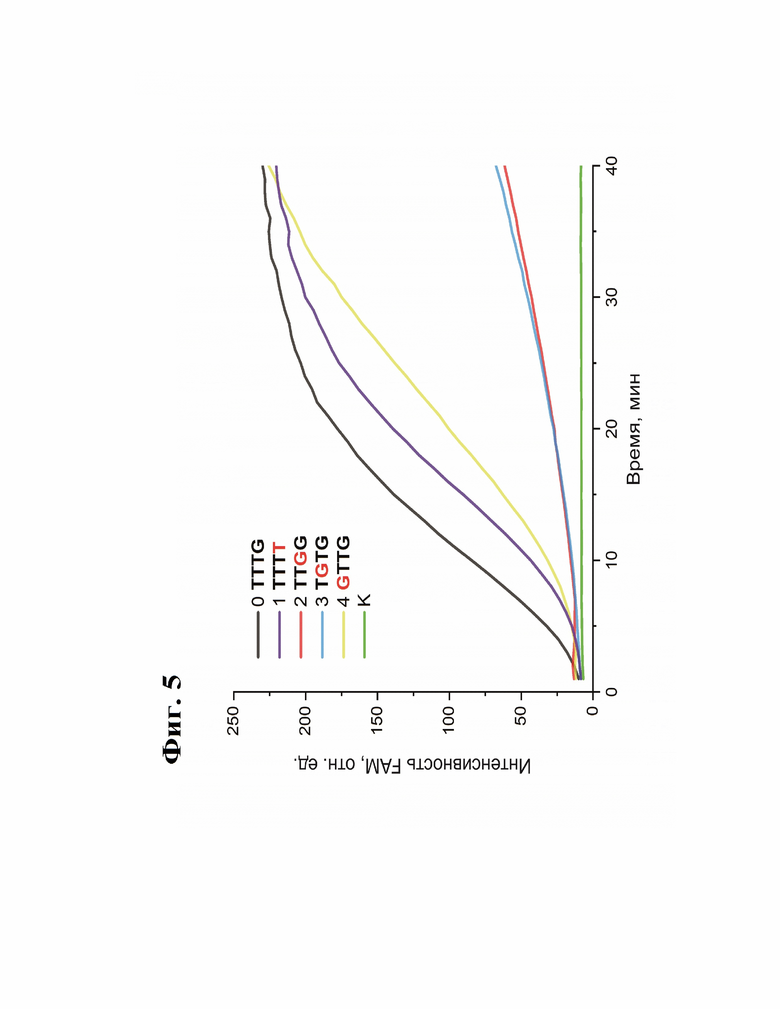
Фиг. 5. Кривые, отображающие динамику изменения интенсивности флуоресценции от времени в процессе расщепления зондов FAM-TTATTATT-BHQ1 рибонуклеопротеиновым комплексом ScCas12a в зависимости от последовательности PAM ДНК мишени.
0 - ДНК-мишень, содержащая правильную последовательность PAM для ScCas12a (5'-TTTG-3').
1-4 - ДНК-мишени, содержащие замены в нуклеотидной последовательности PAM в различных позициях (замены обозначены красным шрифтом).
К - контрольная реакция, не содержащая эффектора ScCas12a.
Подробное раскрытие изобретения
В описании данного изобретения термины «включает» и «включающий» интерпретируются как означающие «включает, помимо всего прочего». Указанные термины не предназначены для того, чтобы их истолковывали как «состоит только из». Если не определено отдельно, технические и научные термины в данной заявке имеют стандартные значения, общепринятые в научной и технической литературе.
Используемый здесь термин «процент гомологии двух последовательностей» эквивалентен термину «процент идентичности двух последовательностей». Идентичность последовательностей определяется на основании референсной последовательности. Алгоритмы для анализа последовательности известны в данной области, например, BLAST (Altschul S. F., Gish W., Miller W., Myers E. W. & Lipman D. J. Basic local alignment search tool // Journal of Molecular Biology. - 1990. - Т. 215. - С. 403-410). Для целей настоящего изобретения для определения уровня идентичности и сходства между нуклеотидными последовательностями и аминокислотными последовательностями может быть использовано сравнение нуклеотидных и аминокислотных последовательностей, производимое с помощью пакета программ BLAST, предоставляемого National Center for Biotechnology Information (http://www.ncbi.nlm.nih.gov/blast) с использованием содержащего разрывы выравнивания со стандартными параметрами. Процент идентичности двух последовательностей определяется числом положений идентичных аминокислот в этих двух последовательностях с учетом числа пробелов и длины каждого пробела, которые необходимо ввести для оптимального сопоставления двух последовательностей путем выравнивания. Процент идентичности равен числу идентичных аминокислот в данных положениях с учетом выравнивания последовательностей, разделенному на общее число положений и умноженному на 100.
Термин «специфически гибридизуется» относится к ассоциации между двумя одноцепочечными молекулами нуклеиновых кислот или в достаточной степени комплементарными последовательностями, что разрешает такую гибридизацию в предопределенных условиях, обычно использующихся в данной области.
Настоящее изобретение охватывает применение белка из организма Sedimentisphaera cyanobacteriorum, гомологичного ранее охарактеризованным белкам Cas12a, для неспецифического внесения разрывов в однонитевые молекулы ДНК в ответ на связывание со строго определенной ДНК-мишенью.
ScCas12a - Cas нуклеаза, найденная в бактериях Sedimentisphaera cyanobacteriorum L21-RPul-D3, выделенных из гиперсоленого озера. CRISPR-Cas система из Sedimentisphaera cyanobacteriorum относится к V-A типу и состоит из CRISPR кассеты, несущей 14 прямых повторов (direct repeats, DR) последовательностью
5'-GTCTAAAGGCCTTATAAAATTTCTACTGTCGTAGAT-3' (SEQ ID NO: 2)
разделенных последовательностями уникальных спейсеров. К CRISPR кассете прилегают гены белков адаптационного модуля Cas2, Cas1, Cas4 и эффекторного Cas12a белка ScCas12a. Пре-крРНК транскрибируется в том же направлении, что и Cas гены (Фиг. 1).
Анализ последовательности локуса ScCas12a позволил предсказать последовательности крРНК:
5'-GUCUAAAGGCCUUAUAAAAUUUCUACUGUCGUAGAUxxxxxxxxxxxxxxxxxxxx-3'
Также показана эффективность расщепления целевой мишени при использовании крРНК с укороченной последовательностью прямого повтора (DR):
5'-AAAUUUCUACUGUCGUAGAUxxxxxxxxxxxxxxxxxxxx-3'
Жирным шрифтом обозначена последовательность DR. Знаками «x» обозначены нуклеотиды вариабельной спейсерной части крРНК.
В настоящем изобретении использовалась крРНК следующей последовательности:
5'-AAAUUUCUACUGUCGUAGAUGUGCUCAAUGAAAGGAGAUA-3' (SEQ ID NO: 3)
Жирным шрифтом обозначена последовательность DR. Красным цветом обозначены нуклеотиды вариабельной спейсерной части крРНК.
PAM, распознаваемый нуклеазой ScCas12a, соответствует следующей формуле:
5'-(T/C)TTN-3' (Фиг. 2).
Для получения рекомбинантного белка ScCas12a его ген был клонирован в плазмиду pET21a-6xHis. В качестве кодирующей ген ДНК, использовалась ДНК, амплифицированная с геномной ДНК Sedimentisphaera cyanobacteriorum L21-RPul-D3, заказанной из коллекции DSMZ (Leibniz Institute DSMZ-German Collection of Microorganisms and Cell Cultures GmbH). Клетки E. coli Rosetta были трансформированы полученной плазмидой pET21a-6xHis-ScCas12a.
5 мл ночной культуры разводили в 500 мл среды LB, и инкубировали при температуре 37°C с равномерным перемешиванием до достижения оптической плотности клеток 0.6 отн. ед. Синтез целевого белка индуцировали добавлением ИПТГ до концентрации 1 мМ, после чего клетки инкубировали при температуре 16°C в течение 16 часов. Затем проводили центрифугирование клеток при относительном ускорении 5000 g в течение 30 минут, полученные осадки клеток замораживали при температуре -20°C.
Осадки размораживали на льду в течение 30 минут, ресуспензировали в 15 мл лизисного буфера (Tris-HCl 40 мМ pH 8, 500 мМ NaCl, β-меркаптоэтанол 1 мМ, имидазол 10 мМ) с добавлением 15 мг лизоцима и снова инкубировали на льду в течение 30 минут. Затем клетки разрушали воздействием ультразвука в течение 30 минут и центрифугировали в течение 60 минут при 16000 g. Полученный супернатант пропускали через фильтр 0.2 мкм и наносили на колонку HisTrap HP 1 mL (GE Healthcare) при помощи FPLC хроматографа AKTA (GE Healthcare) на скорости 0.5 мл/мин. Колонку с нанесенным белком промывали 20 мл лизисного буфера с добавлением 10 мМ имидазола, после чего белок элюировали лизисным буфером с добавлением 300 мМ имидазола.
Затем, фракцию белка, полученную в ходе афинной хроматографии, пропускали через гель-фильтрационную колонку Superdex 200 10/300 GL, уравновешенную следующим буфером: Tris-HCl 40 мМ pH 8, 500 мМ NaCl, 1 мМ DTT. При помощи концентратора Аmicon (с фильтром на 100 кДа) фракции, соответствующие мономерной форме белка ScCas12a, сконцентрировали до 2 мг/мл, после чего очищенный белок хранили при температуре -80°C в буфере, содержащем 10% глицерин.
Для измерения коллатеральной активности ScCas12a использовали зонды - короткие модифицированные олигонуклеотиды с присоединенным флуорофором FAM и гасителем BHQ1, разделенные нуклеотидной последовательностью: FAM-TTATTATT-BHQ1. Флуорофор и гаситель в такой конструкции расположены на небольшом расстоянии, что приводит к эффективной передаче возбуждения от FAM к BHQ1 за счет механизма FRET (Dietrich A., Buschmann V., Müller C. & Sauer M. Fluorescence resonance energy transfer (FRET) and competing processes in donor-acceptor substituted DNA strands: a comparative study of ensemble and single-molecule data // Reviews in Molecular Biotechnology. - 2002. - Т. 82. - №3. - С. 211-231).
Длина волны возбуждения выбирается так, чтобы происходило эффективное возбуждение FAM. При этом в эмиссионном спектре свечение FAM близко к нулю, вследствие передачи возбуждения на BHQ1. При разрезании олигонуклеотидов FAM-TTATTATT-BHQ1 передача возбуждения от FAM к BHQ1 прекращается и в спектре эмиссии появляется интенсивная полоса FAM.
Реакции проводили с использованием ДНК фрагментов, содержащих мишень 5'-gtgctcaatgaaaggagataaggt-3', фланкированную PAM последовательностью 5'-TTTG-3':

Для выявления коллатеральной активности ScCas12a использовали следующие условия.
40 нМ ДНК-мишени, 400 нМ белка ScCas12a, 4 мкМ крРНК, 360 нМ зондов FAM-TTATTATT-BHQ1 в буфере 1x CutSmart (общий объем 40мкл) инкубировали при 37°С в течение 10 мин. В качестве контроля использовался образец без добавления белка ScCas12a. Далее пробы наносили в кюветы и производили измерения с помощью спектрофлуориметра. Спектры эмиссии зондов FAM-TTATTATT-BHQ1, подвергшихся и не подвергшихся расщеплению, при возбуждении излучением с длиной волны 470 нм показаны на Фиг. 3 (кривые 1 и 2, соответственно).
Спектры эмиссии образцов записывались на флуориметре при возбуждении 470 нм в диапазоне 500-600 нм. В спектрах имелась полоса свечения FAM с максимумом на 520 нм. В эксперименте использовались следующие параметры для флуориметра (Carry Eclipce): щели 10 нм (возбуждение), 5 нм (эмиссия), длина волны возбуждения 470 нм, эмиссионный спектр 500-600 нм, скорость сканирования - medium (600 нм/мин), данные через 1 нм, фильтр возбуждения - авто, фильтр регистрации 430-1100 нм, напряжение ФЭУ- medium.
Обработка полученных результатов проводилась следующим образом.
Вводился параметр g, характеризующий интенсивность полосы свечения FAM (усреднение по интервалу 8 нм):
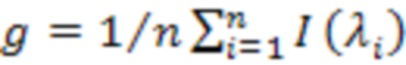 (1), где
(1), где
n - количество измерений интенсивности в диапазоне 516-524 нм. Если образец содержит только олигонуклеотиды, не подвергшиеся расщеплению, g0 = 8.8 ± 0.8, при нуклеотидах, полностью подвергшихся расщеплению, gf = 220 ± 10. Данные значения зависят от типа флуориметра и концентрации модифицированных олигонуклеотидов в образце, и должны определяться экспериментально.
Спектр флуоресценции образца является суммой интенсивностей олигонуклеотидов, подвергшихся и не подвергшихся расщеплению, поэтому от параметра g можно перейти к относительному количеству нуклеотидов, подвергшихся расщеплению (α):
 (2).
(2).
Зависимость α от g показана на Фиг. 4.
Таким образом, по измеренному эмиссионному спектру образца с помощью формулы (1) определялся параметр g. Далее с помощью формулы (2) определялось относительное количество зондов, подвергшихся расщеплению. На основании анализа можно судить что все зонды FAM-TTATTATT-BHQ1, находящиеся в реакции с рибонуклеинопротеновым комплексом ScCas12a, активированным ДНК мишенью, были расщеплены, и детектировался максимум возможного сигнала. Из чего следует, что рибонуклеинопротеновый комплекс ScCas12a обладает высокоэффективной коллатеральной активностью, которая и представляет суть изобретения.
Нижеследующие примеры осуществления способа приведены в целях раскрытия характеристик настоящего изобретения и их не следует рассматривать как каким-либо образом ограничивающие объем изобретения.
Пример 1. Тестирование коллатеральной активности белка ScCas12a при расщеплении ДНК мишени, фланкированной различными последовательностями PAM.
Соответствие последовательности PAM 5'-(T/C)TTN-3' является обязательным требованием рибонуклеинопротеинового комплекса ScCas12a для осуществления специфического расщепления ДНК мишени, что ведет к конформационным преобразованиям комплекса ScCas12a для активации коллатеральной активности. Таким образом, изобретение может быть использовано для детекции одиночных и комплексных нуклеотидных замен в том числе инсерции и делеции в области PAM интересующей ДНК мишени. Тестирование чувствительности к одиночным заменам в области PAM проводили с использованием спектрофлуориметра, анализируя динамику коллатеральной активности ScCas12a в режиме реального времени.
Реакции проводили с использованием ДНК фрагментов, содержащих мишень 5'-gtgctcaatgaaaggagataaggt-3', фланкированную PAM последовательностями 5'-TTTG-3', 5'-TTTT-3', 5'-TTGG-3', 5'-TGTG-3' или 5'-GTTG-3':

На Фиг. 5 представлены графики зависимости интенсивности флуоресценции зондов, подвергшихся расщеплению, от времени для мишеней с одиночными заменами в области последовательности PAM. Реакции проводили в следующих условиях:
4 нМ ДНК-мишени, 8 нМ белка ScCas12a, 80 нМ крРНК, 360 нМ зондов FAM-TTATTATT-BHQ1 в буфере с CutSmart инкубировались при 37°С непосредственно в кювете спектрофлуориметра в режиме регистрации сигнала. Кривые 2 и 3 демонстрируют значительное понижение коллатеральной активности в случае замены критически важных позиций в области PAM (Фиг. 5). Таким образом показана возможность детектирования изменения в нуклеотидной последовательности PAM, что может быть использовано, например, для детекции мутаций возбудителей инфекционных заболеваний.
Пример 2. Описанная в настоящем изобретении нуклеаза ScCas12a в комплексе с крРНК может быть использована для детектирования определенных нуклеиновых кислот.
Так, например, нуклеаза, относящаяся к типу V CRISPR-Cas систем, используется в основе платформы DETECTR (DNA Endonuclease Targeted CRISPR Trans Reporter) вместе с направляющей РНК, специфичной к нуклеиновой кислоте, и флуоресцентной репортерной молекулой. Данная платформа успешно использовалась для выявления и генотипирования вируса папилломы человека (Chen J. S., Ma E., Harrington L.B., Da Costa M., Tian X., Palefsky J. M. & Doudna J. A. CRISPR-Cas12a target binding unleashes indiscriminate single-stranded DNase activity // Science. - 2018. - Т. 360. - №6387. - С. 436-439). Недавно также вышла ее модификация для выявления РНК вируса SARS-CoV-2 (Broughton J. P., Deng X., Yu G., Fasching C. L., Servellita V., Singh J., Miao X., Streithorst J. A., Granados A., Sotomayor-Gonzalez A., Zorn K., Gopez A., Hsu E., Gu W., Miller S., Pan C.-Y., Guevara H., Wadford D. A., Chen J. S. & Chiu C. Y. CRISPR-Cas12-based detection of SARS-CoV-2 // Nature biotechnology. - 2020. - Т. 38. - №7. - С. 870-874).
ScCas12a - новая Cas нуклеаза, значительно отличающаяся по аминокислотной последовательности от описанных ранее Cas12a нуклеаз и имеющая простой в использовании PAM. Белок ScCas12a расщепляет с высокой эффективностью ДНК-мишени, в том числе и при 37°С и обладает коллатеральной активность.
Несмотря на то, что изобретение описано со ссылкой на раскрываемые варианты воплощения, для специалистов в данной области должно быть очевидно, что конкретные подробно описанные случаи приведены лишь в целях иллюстрирования настоящего изобретения, и их не следует рассматривать как каким-либо образом ограничивающие объем изобретения. Должно быть понятно, что возможно осуществление различных модификаций без отступления от сути настоящего изобретения.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing originalFreeTextLanguageCode="ru"
dtdVersion="V1_3" fileName="Средство обнаружения нуклеиновых кислот
на основе ScCas12a белка из бактерии Sedimentisphaera
cyanobacteriorum.xml" softwareName="WIPO Sequence"
softwareVersion="2.1.2" productionDate="2023-05-10">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>-</ApplicationNumberText>
<FilingDate></FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>-</ApplicantFileReference>
<ApplicantName languageCode="ru">Федеральное государственное
бюджетное учреждение науки Институт биологии гена Российской академии
наук </ApplicantName>
<ApplicantNameLatin>Institute of Gene Biology Russian Academy of
Sciences</ApplicantNameLatin>
<InventionTitle languageCode="ru">Средство обнаружения нуклеиновых
кислот на основе ScCas12a белка из бактерии Sedimentisphaera
cyanobacteriorum</InventionTitle>
<SequenceTotalQuantity>5</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>1310</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..1310</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Sedimentisphaera
cyanobacteriorum</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MKEFTNQYSLTKTLRFELRPVGETAEKIEDFKSGGLKQTVEKDRERTEA
YKQLKEVIDSYHRDFIEQAFARQQTLSEEDFKQTYQLYKEAQKEKDGETLTKQYEHLRKKIAAMFSKATK
EWAVMGENNELIGKNKESKLYQWLEKNYRAGRIEKEEFDHNAGLIEYFEKFSTYFVGFDKNRANMYSKEA
KATAISFRTINENMVKHFDNCQRLEKIKSKYPDLAEELKDFEEFFKPSYFINCMNQSGIDYYNISAIGGK
DEKDQKANMKINLFTQKNHLKGSDKPPFFAKLYKQILSDREKSVVIDEFEKDSELTEALKNVFSKDGLIN
EEFFTKLKSALENFMLPEYQGQLYIRNAFLTKISANIWGSGSWGIIKDAVTQAAENNFTRKSDKEKYAKK
DFYSIAELQQAIDEYIPTLENGVQNASLIEYFRKMNYKPRGSEEDAGLIEEINNNLRQAGIVLNQAELGS
GKQREENIEKIKNLLDSVLNLERFLKPLYLEKEKMRPKAANLNKDFCESFDPLYEKLKTFFKLYNKVRNY
ATKKPYSKDKFKINFDTATLLYGWSLDKETANLSVIFRKREKFYLGIINRYNSQIFNYKIAGSESEKGLE
RKRSLQQKVLAEEGEDYFEKMVYHLLLGASKTIPKCSTQLKEVKAHFQKSSEDYIIQSKSFAKSLTLTKE
IFDLNNLRYNTETGEISSELSDTYPKKFQKGYLTQTGDVSGYKTALHKWIDFCKEFLRCYRNTEIFTFHF
KDTKEYESLDEFLKEVDSSGYEISFDKIKASYINEKVNAGELYLFEIYNKDFSEYSKGKPNLHTIYWKSL
FETQNLLDKTAKLNGKAEIFFRPRSIKHNDKIIHRAGETLKNKNPLNEKPSSRFDYDITKDRRFTKDKFF
LHCPITLNFKQDKPVRFNEQVNLYLKDNPDVNIIGIDRGERHLLYYTLINQNGEILQQGSLNRIGEEESR
PTDYHRLLDEREKQRQQARETWKAVEGIKDLKAGYLSRVVHKLAGLMVQNNAIVVLEDLNKGFKRGRFAV
EKQVYQNFEKALIQKLNYLVFKEVNSKDAPGHYLKAYQLTAPFISFEKLGTQSGFLFYVRAWNTSKIDPA
TGFTDQIKPKYKNQKQAKDFMSSFDSVRYNRKENYFEFEADFEKLAQKPKGRTRWTICSYGQERYSYSPK
ERKFVKHNVTQNLAELFNSEGISFDSGQCFKDEILKVEDASFFKSIIFNLRLLLKLRHTCKNAEIERDFI
ISPVKGNNSSFFDSRIAEQENITSIPQNADANGAYNIALKGLMNLHNISKDGKAKLIKDEDWIEFVQKRK
F</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>36</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..36</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Sedimentisphaera
cyanobacteriorum</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtctaaaggccttataaaatttctactgtcgtagat</INSDSeq_seq
uence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>40</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..40</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q3">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aaatttctactgtcgtagatgtgctcaatgaaaggagata</INSDSeq
_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>374</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..374</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aataccagagataagagagtaggctggtagatggagttgggtttggtgc
tcaatgaaaggagataaggtccttgaattgcagtatctagcctcttctaagacaggttacgtgatgtaga
tcctattttaacatgctctttctttgtgtttgcagggagtcgacgagttgaagatgaagcccagagcgga
gtgctgttctcccaagttctggttggtgttggccgtcctggccgtgtcaggcagcagagctcgttctcag
aagagcccccccagcattggcattgctgtcatcctcgtgggcacttccgacgaggtggccatcaaggatg
cccacgagaaagatgatttccaccatctctccgtggtaccccggg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>374</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..374</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q5">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aataccagagataagagagtaggctggtagatggagttgggnnnngtgc
tcaatgaaaggagataaggtccttgaattgcagtatctagcctcttctaagacaggttacgtgatgtaga
tcctattttaacatgctctttctttgtgtttgcagggagtcgacgagttgaagatgaagcccagagcgga
gtgctgttctcccaagttctggttggtgttggccgtcctggccgtgtcaggcagcagagctcgttctcag
aagagcccccccagcattggcattgctgtcatcctcgtgggcacttccgacgaggtggccatcaaggatg
cccacgagaaagatgatttccaccatctctccgtggtaccccggg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Средство разрезания ДНК на основе ScCas12a белка из бактерии Sedimentisphaera cyanobacteriorum | 2022 |
|
RU2791447C1 |
| Средство разрезания ДНК на основе Cas9 белка из бактерии Streptococcus uberis NCTC3858 | 2022 |
|
RU2788197C1 |
| Средство разрезания ДНК на основе Cas9 белка из бактерии Capnocytophaga ochracea | 2021 |
|
RU2778156C1 |
| Средство разрезания двунитевой ДНК с помощью Cas12d белка из Katanobacteria и гибридной РНК, полученной путем слияния направляющей CRISPR РНК и scout РНК | 2020 |
|
RU2771626C1 |
| СРЕДСТВО РАЗРЕЗАНИЯ ДНК НА ОСНОВЕ CAS9 БЕЛКА ИЗ БАКТЕРИИ DEMEQUINA SEDIMINICOLA | 2019 |
|
RU2722933C1 |
| НУКЛЕАЗА CPF1 ИЗ БАКТЕРИИ Ruminococcus bromii, МОЛЕКУЛА ДНК ИЛИ РНК, КОДИРУЮЩАЯ НУКЛЕАЗУ, ВЕКТОР, СОДЕРЖАЩИЙ УКАЗАННУЮ МОЛЕКУЛУ ДНК, СИСТЕМА CRISPR/CPF1, СОДЕРЖАЩАЯ УКАЗАННУЮ НУКЛЕАЗУ И НАПРАВЛЯЮЩУЮ РНК, КЛЕТКА-ХОЗЯИН ДЛЯ ПОЛУЧЕНИЯ НУКЛЕАЗЫ CPF1, СПОСОБ ПОЛУЧЕНИЯ НУКЛЕАЗЫ CPF1 И ЕЕ ПРИМЕНЕНИЕ | 2022 |
|
RU2816876C1 |
| СРЕДСТВО РАЗРЕЗАНИЯ ДНК НА ОСНОВЕ CAS9 БЕЛКА ИЗ DEFLUVIIMONAS SP. | 2018 |
|
RU2712492C1 |
| Средство разрезания ДНК на основе Cas9 белка из биотехнологически значимой бактерии Clostridium cellulolyticum | 2018 |
|
RU2712497C1 |
| СРЕДСТВО РАЗРЕЗАНИЯ ДНК НА ОСНОВЕ CAS9 БЕЛКА ИЗ БАКТЕРИИ PASTEURELLA PNEUMOTROPICA | 2019 |
|
RU2722934C1 |
| ПРИМЕНЕНИЕ CAS9 БЕЛКА ИЗ БАКТЕРИИ PASTEURELLA PNEUMOTROPICA ДЛЯ МОДИФИКАЦИИ ГЕНОМНОЙ ДНК В КЛЕТКАХ | 2019 |
|
RU2724470C1 |
Настоящее изобретение относится к области молекулярной биологии. Описано применение белка, содержащего аминокислотную последовательность SEQ ID NO: 1, для неспецифического расщепления одноцепочечных молекул ДНК. Также описан способ детектирования нуклеиновых кислот живых организмов, включающий применение указанного белка. Техническим результатом настоящего изобретения является создание нового инструмента определения специфических последовательностей нуклеиновых кислот на основе коллатеральной активности нуклеазы ScCas12a. 4 н. и 1 з.п. ф-лы, 5 ил., 2 пр.
1. Применение белка, содержащего аминокислотную последовательность SEQ ID NO: 1, для неспецифического расщепления одноцепочечных молекул ДНК после связывания с целевым участком на двуцепочечной молекуле ДНК, расположенным после нуклеотидной последовательности 5'-TTTN-3' в указанной двуцепочечной молекуле ДНК.
2. Применение белка, содержащего аминокислотную последовательность SEQ ID NO: 1, для неспецифического расщепления одноцепочечных молекул ДНК после связывания с целевым участком на двуцепочечной молекуле ДНК, расположенным после нуклеотидной последовательности 5'-CTTN-3' в указанной двуцепочечной молекуле ДНК.
3. Применение белка по п. 1, где белок содержит аминокислотную последовательность SEQ ID NO: 1, для неспецифического расщепления одноцепочечных молекул ДНК после связывания с целевым участком на двуцепочечной молекуле ДНК, расположенным после нуклеотидной последовательности, обладающей единичными заменами в сравнении с консенсусом 5'-(T/C)TTN-3', в указанной двуцепочечной молекуле ДНК.
4. Способ детектирования нуклеиновых кислот живых организмов, включающий использование: а) белка, содержащего аминокислотную последовательность SEQ ID NO: 1, б) направляющей РНК, содержащей последовательность, образующую дуплекс с нуклеотидной последовательностью целевой молекулы ДНК, непосредственно примыкающей к нуклеотидной последовательности 5'-TTTN-3', в) зондов, включающих короткие модифицированные олигонуклеотиды с присоединенным флуорофором FAM и гасителем BHQ1, разделенные нуклеотидной последовательностью: FAM-TTATTATT-BHQ1; при этом взаимодействие указанного белка с направляющей РНК и нуклеотидной последовательностью 5'-TTTN-3' приводит к образованию двунитевого разрыва в последовательности целевой ДНК, непосредственно примыкающей к последовательности 5'-TTTN-3', и неспецифическому расщеплению зондов.
5. Способ детектирования нуклеиновых кислот живых организмов, включающий использование: а) белка, содержащего аминокислотную последовательность SEQ ID NO: 1, б) направляющей РНК, содержащей последовательность, образующую дуплекс с нуклеотидной последовательностью целевой молекулы ДНК, непосредственно примыкающей к нуклеотидной последовательности 5'-CTTN-3', в) зонды, включающие короткие модифицированные олигонуклеотиды с присоединенным флуорофором FAM и гасителем BHQ1, разделенные нуклеотидной последовательностью: FAM-TTATTATT-BHQ1; при этом взаимодействие указанного белка с направляющей РНК и нуклеотидной последовательностью 5'-CTTN-3' приводит к образованию двунитевого разрыва в последовательности целевой ДНК, непосредственно примыкающей к последовательности 5'-CTTN-3', и неспецифическому расщеплению зондов.
| CN 109666662 A, 23.04.2019 | |||
| СРЕДСТВО РАЗРЕЗАНИЯ ДНК НА ОСНОВЕ CAS9 БЕЛКА ИЗ DEFLUVIIMONAS SP. | 2018 |
|
RU2712492C1 |
| ASTRID WENINGER et al | |||
| Разборный с внутренней печью кипятильник | 1922 |
|
SU9A1 |
| Elsevier | |||
| Упругая металлическая шина для велосипедных колес | 1921 |
|
SU235A1 |
Авторы
Даты
2024-06-03—Публикация
2023-03-30—Подача