Изобретение относится к медицине, а именно к молекулярной биологии, онкологии, и может быть использовано для определения статуса генов IDH1 и IDH2 у пациентов с диагнозом глиальная опухоль головного мозга.
Опухоли головного мозга занимают значительную часть в общей структуре онкологических заболеваний в РФ: 3,7% у мужчин и 3,9% у женщин. В 2017 году смертность от ЗНО в России составила 617177 случаев, из них 8844 - опухоли головного мозга и др. отделов ЦНС (см. под редакцией А.Д. Каприна, В.В. Старинского, Г.В. Петровой // Злокачественные новообразования в России в 2017 году (см. заболеваемость и смертность), Москва, 2018).
Глиомы - нейроэпителиальные опухоли, возникающие из глиальных клеток мозга и занимающие основную часть всех первичных опухолей головного мозга. До пересмотра классификации ВОЗ в 2016 году первичные глиальные опухоли классифицировались по 4м степеням злокачественности исключительно на основе гистологических, фенотипических и морфологических признаков.
В 2016 году классификация ВОЗ изменена и включает как гистологические, так и молекулярно-генетические биомаркеры глиальных опухолей. Молекулярные маркеры стали неотъемлемой частью классификации глиальных опухолей. Основные маркеры, регулярно оцениваемые в клинической практике - наличие мутаций в генах изоцитратдегидрогеназы (IDH) (мутантный или дикий тип) и коделеции 1p/19q (совместная делеция или ее отсутствие). На основании этих маркеров, классификация ВОЗ определила диагностику олигодендроглиом (IDH1/2 mut, 1p/19q коделеция) и астроцитом (IDH1/2 mut, без 1p/19q коделеции), для которых также характерна делеция ATRX и мутация P53 (см. Louis D. N. et al. The 2016 World Health Organization classification of tumors of the central nervous system: a summary //Acta neuropathologica. - 2016. - Т. 131. - №. 6. - С. 803-820).
Сочетание гистологических и молекулярных маркеров позволит достичь более воспроизводимой постановки диагноза, и более точного прогнозирования исхода заболевания. Молекулярная классификация связана с выживаемостью у пациентов с глиомами. Например, пациенты с мутантным статусом IDH1/2 и метилированием MGMT имеют лучший прогноз (медиана общей выживаемости около 36 месяцев); пациенты с мутацией IDH1/2 или метилированием MGMT показывают промежуточный прогноз (медиана выживаемости около 14 месяцев); и пациенты с IDH1/2 дикого типа и неметилированным промотором гена MGMT имеют худший прогноз (медиана общей выживаемости около 9 месяцев) (см. Molinari E., Curran O. E., Grant R. Clinical importance of molecular markers of adult diffuse glioma //Practical neurology. - 2019. - С. practneurol-2018-002116.).
Гены IDH1 и IDH2 кодируют цитоплазматическую и митохондриальную изоцитратдегидрогеназу соответственно. Изоцитратдегидрогеназа катализирует третью реакцию цикла Кребса - преобразование изоцитрата в α-кетоглутарат (с восстановлением НАД). Частота встречаемости мутаций в IDH1 - 39,84%, среди которых идентифицируют R132H (наиболее встречаемая), R132C, R132G, R132S, R132L. Частота встречаемости мутаций в гене IDH2 - 2,78%, среди которых наиболее часто встречаются R140Q и R172K.
Новая классификация первичных опухолей головного мозга требует диагностики ряда молекулярных маркеров, в том числе статус IDH1/2, для диагностики и определения стратегии лечения.
Анализ литературных источников показал наличие близких по тематике изобретений:
1) «Клинические, иммуногистохимические и молекулярно-генетические факторы прогноза у больных c глиобластомой.» (doi: 10.17116/patol201678410-19). Сущностью способа является проведение иммуногистохимического окрашивания мутантных форм IDH1 с использованием антител к R 132.
2) «Феномен длительной выживаемости пациентов с глиобластомами.
Часть I: роль клинико-демографических факторов и мутации IDH1 (R 132 H)» (doi: 10.17116/neiro20178135-16). Сущностью способа так же заключается в проведении иммуногистохимического окрашивания мутантных форм IDH 1 с использованием антител к R 132.
Описанные подходы используют в анализе способы принципиально отличающиеся от нашего; обладающие меньшей чувствительностью, чем предлагаемая нами тест-система. В приведенных работах определяют только R132H в гене IDH1, из семи клинически значимых мутаций.
Анализ патентных источников (www.fips.ru) показал отсутствие действующих патентов и заявок на патент тест-системы для определения статуса генов IDH ½ в тканях глиальной опухоли на базе молекулярно-генетических методов.
Тест-система для определения статуса генов IDH ½ в тканях глиальных опухолей является новой, так как использует комбинацию двух технологий HRM (High Resolution Melt) и прямого секвенирования по Сэнгеру, а также высокоспецифичные синтетические олигонуклеотиды и алгоритм обработки данных, повышающие экономическую и техническую эффективность анализа статуса генов IDH ½.
Техническим результатом изобретения является создание и внедрение новой, простой в исполнении, не дорогостоящей и точной тест-системы с уникальными высокоспецифичными последовательностями синтетических олигонуклеотидов (праймеров) для определения статуса генов IDH ½ в тканях глиальной опухоли.
Сущность тест-системы заключается в том, что геномную ДНК экстрагируют из образцов опухолевой ткани головного мозга, и, используя полученный препарат ДНК в качестве матрицы, проводят HRM амплификацию с высокоспецифичными праймерами для генов IDH1 и IDH2, анализируют кривые плавления - сравнивают кривые плавления ампликонов образцов (HRMs) с кривыми плавления позитивного (с наличием мутации, HRMm) и негативного контроля (без мутации, HRMd). При соответствии кривых плавления HRMs кривым плавления HRMm (0.03>difRFU>-0.03) проводят секвенирование по Сэнгеру исследуемых образцов для валидации результата (подтверждения наличия мутации), при соответствии кривых плавления HRMs кривым плавления HRMd (0.03>difRFU>-0.03) образцы считаются несущими референсный (дикий) вариант генов IDH1 и IDH2, при несоответствии кривых плавления HRMs ни HRMm, ни HRMd проводят секвенирование по Сэнгеру исследуемых образцов для определения наличия или отсутствия мутации.
Для лучшего понимания способа приводим фигуры.
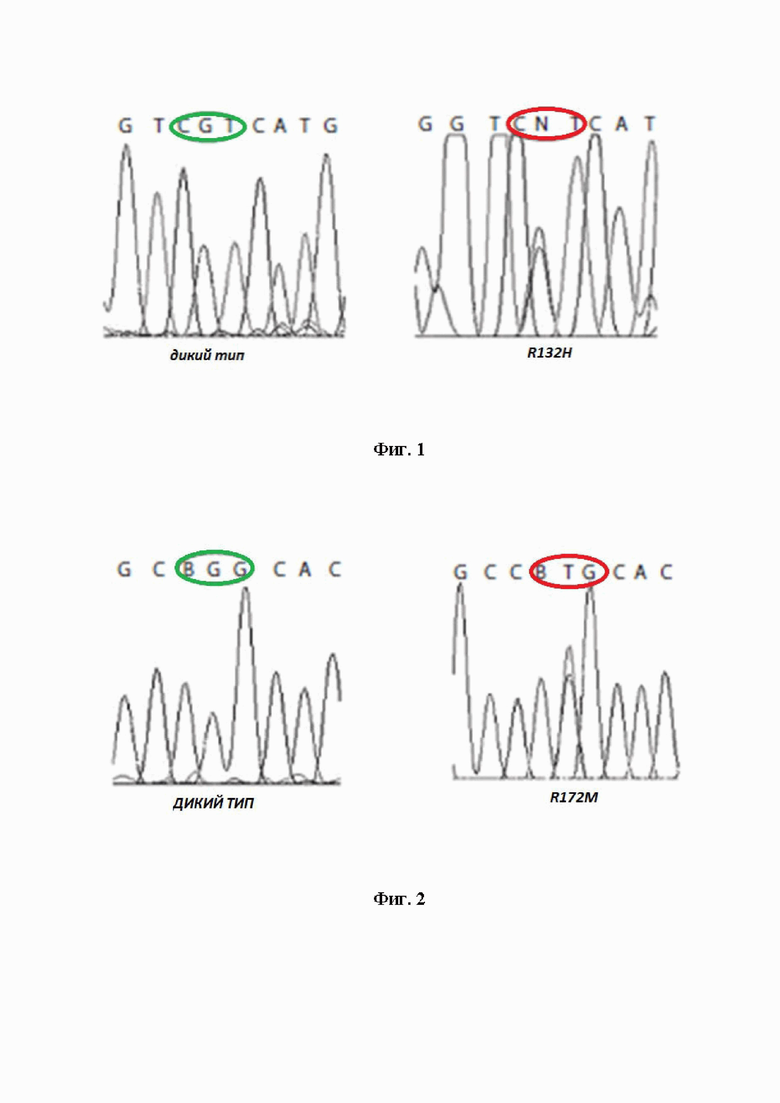
Фигура 1. Секвенированный участок гена IDH1 дикого типа и с мутацией R132H.
Фигура 2. Секвенированный участок гена IDH2 дикого типа и с мутацией R172M.
Заявленный анализ основан на определении мутаций в генах IDH1 и IDH2 в опухолевой ткани с использованием HRM и секвенирования по Сэнгеру.
Заявленная тест-система включает следующие приёмы: выделение тотальной ДНК из тканевых проб или из парафиновых блоков с помощью подходящего набора для экстракции; определение наличия мутаций в генетических локусах IDH1/2 методом ПЦР-РВ c HRM в присутствии красителя EVA-Green и специфичных праймеров на матрице выделенной ДНК; анализ первичных данных с помощью программного продукта амплификатора; в случае необходимости дальнейшее секвенирование по Сэнгеру, анализ данных секвенирования.
Заявленная тест-система, рассчитанная на 50 реакций, включает:
1.Смесь для ПЦР-РВ реакции (Mix_PCR: 10Х ПЦР-буффер с 20 mM MgCl и 12,5 mM dNTPs ) - 1500 мкл.
2. ДНК-полимеразу Thermus aquaticus 5ед/мкл (Taq) - 50 мкл.
3. Смесь прямого и обратного HRM праймеров с концентрацией 10 мкМ каждого для локуса IDH1 -500 мкл (смесь включает воду для ПЦР - реакции).
4. Смесь прямого и обратного HRM праймеров с концентрацией 10 мкМ каждого для гена IDH2 - 500 мкл (смесь включает воду для ПЦР - реакции)
5. Негативный контрольный образец без мутации IDH1 и IDH2 (HRMd) - 1000 мкл (смесь включает воду для ПЦР - реакции).
6. Положительный контрольный образец с мутацией IDH1 (HRMm R132) - 500 мкл.
7. Положительный контрольный образец с мутацией IDH2 (HRMm R140Q) - 500 мкл.
8. Положительный контрольный образец с мутацией IDH2 (HRMm R172K) - 500 мкл.
9. Смесь прямого и обратного праймеров для секвенирования с концентрацией 10 мкМ каждого для локуса IDH1 -500 мкл (смесь включает воду для ПЦР - реакции).
10. Смесь прямого и обратного праймеров для секвенирования с концентрацией 10 мкМ каждого для гена IDH2 - 500 мкл (смесь включает воду для ПЦР - реакции).
Для тест-системы были разработаны специфичные олигонуклеотидные прямые и обратные праймеры для локусов IDH1 и IDH2. Дизайн специфичных олигонуклеотидных праймеров (см. таблица 1) осуществлялся с использованием референсных последовательностей NCBI GenBank.
Таблица 1
Тест-система для определения статуса генов IDH ½ в тканях глиальных опухолей
ACATTATTGCCAACATGA(SeqID 2)
AGCCAGTGTTGAAAACCACAG(SeqID 4)
AAGTCTGTGGCCTTGTACTGC(SeqID 6)
CCAAGCCAGCCTCACCTC(SeqID 8)
Для доказательства функциональности предлагаемой тест-системы приводится 3 выписки из историй болезни.
1) Больная Г., 47 лет, госпитализирована в апреле 2018 г. в отделение нейроонкологии ФГБУ «НМИЦ онкологии» Минздрава России с диагнозом диффузная глиальная опухоль лобных долей с вовлечением передних отделов мозолистого тела. По данным ИГХ анапластическая астроцитома, Grade 3.
Результаты молекулярного анализа образца ткани опухоли (операционный биоптат): кривые плавления IDH2 (HRM) образца соответствовали кривым плавления дикого типа (HRMd) (difRFU=-0,01); кривые плавления IDH1 (HRM) образца соответствовали кривым плавления мутантного типа (HRMm) (difRFU=0,01), было проведено секвенирование по Сэнгеру и подтверждено наличие мутации в гене IDH1 - R132H (см. Фиг. 1).
2) Больной Т., 40 лет, госпитализирован в феврале 2019 г. в отделение нейроонкологии ФГБУ «НМИЦ онкологии» Минздрава России с диагнозом глиальная диффузная инфильтративная опухоль правой лобной и височной долей с прорастанием сосудов Сильвиева пакета с ростом в мозолистое тело. По данным ИГХ - астроцитома, Grade 2.
Результаты молекулярного анализа образца ткани опухоли (операционный биоптат): кривые плавления IDH1 (HRM) образца соответствовали кривым плавления дикого типа (HRMd) (difRFU=0,02); кривые плавления IDH2 образца (HRMs) не соответствовали ни кривым плавления позитивного (с наличием мутации, HRMm) контроля, ни кривым плавления негативного контроля (без мутации, HRMd), было проведено секвенирование по Сэнгеру и выявлена мутация в гене IDH2 - R172M (см. Фиг. 2).
3) Больной Д., 33 года, госпитализирован в апреле 2019 г. в отделение нейроонкологии ФГБУ «НМИЦ онкологии» МЗ РФ с диагнозом глиальная опухоль правой лобной доли с распространением на мозолистое тело. По данным ИГХ - глиобластома, Grade 4.Результаты молекулярного анализа образца ткани опухоли (операционный биоптат): кривые плавления образца (HRMs) соответствовали кривым плавления негативного контроля (без мутации) HRMd (0.01>difRFU>-0.01), соответственно образец считали несущим референсный (дикий) вариант генов IDH1 и IDH2. С помощью предлагаемой тест-системы были проанализированы биологические образцы 150 пациентов и выявлено 25 мутаций в гене IDH1 и 2 мутации в гене IDH2.
Технико-экономическая эффективность изобретения. «Тест-система для определения статуса генов IDH ½ в тканях глиальной опухоли» позволяет с высокой точностью определять наличие мутаций в генах IDH1/2. Заявляемая тест-система, включает разработанные нами высокоспецифичные праймеры и является экономически оправданной для уточнения диагноза и особенностей течения заболевания и дает возможность скорректировать тактику лечения, осуществляется в условиях стандартной лаборатории молекулярной биологии, обладает высокой чувствительностью и специфичностью, осуществление анализа возможно с операционными биоптатами и парафиновыми блоками (FFPE-блоки), занимает не более 10 часов (с учетом подготовительных этапов).
--->
Перечень последовательностей
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="Тест-система для
определения статуса генов IDH ½ в тканях глиальных
опухолей_последовательности.xml" softwareName="WIPO Sequence"
softwareVersion="2.3.0" productionDate="2024-04-18">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2023125038/10</ApplicationNumberText>
<FilingDate>2023-09-29</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>055187</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2023125038/10(055187)</ApplicationNumberText>
<FilingDate>2023-09-29</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">Федеральное государственное
бюджетное учреждение "Национальный медицинский исследовательский
центр онкологии" Министерства здравоохранения Российской
Федерации</ApplicantName>
<ApplicantNameLatin>Natsional'nyy meditsinskiy
issledovatel'skiy tsentr onkologii</ApplicantNameLatin>
<InventorName languageCode="ru">Кутилин Денис
Сергеевич</InventorName>
<InventorNameLatin>Kutilin Denis</InventorNameLatin>
<InventionTitle languageCode="ru">Тест-система для определения
статуса генов IDH ½ в тканях глиальных опухолей</InventionTitle>
<SequenceTotalQuantity>8</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cttcagagaagccattatc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>acattattgccaacatga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caacgaccaagtcaccaagg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agccagtgttgaaaaccacag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aacatcccacgcctagtcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aagtctgtggccttgtactgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cagagcccacacatttgcac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q16">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccaagccagcctcacctc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ анализа соматических мутаций в генах IDH1 и IDH2 с использованием LNA-блокирующей ПЦР и гибридизации с биологическим микрочипом | 2024 |
|
RU2839291C1 |
| Набор высокоспецифичных олигонуклеотидных праймеров для детекции генетических вариантов методом высокопроизводительного секвенирования ДНК | 2024 |
|
RU2833429C1 |
| Способ диагностики инвазивного кандидоза и видовой идентификации его основных возбудителей методом ПЦР с гибридизационно-флуоресцентной детекцией в режиме реального времени | 2022 |
|
RU2809386C1 |
| Малоинвазивный способ дифференциальной ранней диагностики гепатоцеллюлярной карциномы и жирового гепатоза | 2023 |
|
RU2822224C1 |
| Мультиплексный набор маркеров как инструмент для разработки генетического тестирования подверженности к шизофрении, алкоголизму и связи с особенностями личности в молодом и умственными способностями в пожилом возрасте | 2022 |
|
RU2819816C1 |
| Способ генотипирования полиморфного локуса rs139794067 (G>T) гена MYL3 | 2025 |
|
RU2837876C1 |
| Способ идентификации SNP-генотипов возбудителя чумы средневекового биовара филогенетических ветвей 2.MED1 и 2.MED4 из очагов Северного и Северо-Западного Прикаспия методом секвенирования по Сэнгеру | 2024 |
|
RU2831366C1 |
| Способ генотипирования однонуклеотидного варианта rs2304277 (G>A) гена OGG1 человека методом полимеразно-цепной реакции в режиме реального времени | 2024 |
|
RU2836047C1 |
| Способ повышения физической выносливости в эксперименте на дисферлин-дефицитных мышах | 2023 |
|
RU2833672C1 |
| Способ генотипирования однонуклеотидного варианта rs3219493 (C>G) гена MUTYH человека методом полимеразно-цепной реакции в режиме реального времени | 2024 |
|
RU2835214C1 |
Изобретение относится к медицине, а именно к молекулярной биологии, онкологии, и может быть использовано для определения статуса генов IDH1 и IDH2 у пациентов с диагнозом «глиальная опухоль головного мозга». Заявленная тест-система содержит реагенты для ПЦР амплификации ДНК - смесь для ПЦР, ДНК-полимеразу Thermus aquaticus, положительные и негативные контрольные образцы, высокоспецифичные праймеры для анализа статуса генов IDH1 и IDH2 методами HRM и секвенирования по Сэнгеру. Тест-система обладает высокой чувствительностью и специфичностью, её использование возможно с операционным биопсийным материалом и парафиновыми блоками (FFPE-блоки); регистрацию результатов производят однократно в конце исследования, занимающего не более 10 часов. Тест-система позволяет осуществить исследование генов IDH 1/2 на наличие мутаций, что дает возможность персонифицировать тактику лечения при глиальных опухолях головного мозга. 2 ил., 1 табл.
Тест-система для определения статуса генов IDH ½ в тканях глиальных опухолей, включающая реагенты для получения ампликонов на матрице ДНК, отличающаяся тем, что используют высокоспецифичные праймеры, имеющие последовательности SEQ ID NO: 1,SEQ ID NO: 2, SEQ ID NO: 3 и SEQ ID NO: 4 для гена IDH1 и SEQ ID NO: 5, SEQ ID NO: 6, SEQ ID NO: 7 и SEQ ID NO: 8 для гена IDH2, анализируют первичные данные и сравнивают кривые плавления ампликонов образцов - HRMs с кривыми плавления позитивного - с наличием мутации, HRMm и негативного контроля - без мутации, HRMd, при соответствии кривых плавления HRMs кривым плавления HRMm: 0.03>difRFU>-0.03 проводят секвенирование по Сэнгеру исследуемых образцов для подтверждения наличия мутации, при соответствии кривых плавления HRMs кривым плавления HRMd: 0.03>difRFU>-0.03 образцы считаются несущими референсный вариант генов IDH1 и IDH2, при несоответствии кривых плавления HRMs, ни HRMm, ни HRMd проводят секвенирование по Сэнгеру исследуемых образцов для определения наличия или отсутствия мутации.
| CN102925579 A, 13.02.2013 | |||
| WO2017129662 A1, 03.08.2017 | |||
| GONDIM D.D | |||
| et al | |||
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
| Прибор с двумя призмами | 1917 |
|
SU27A1 |
| Печь-кухня, могущая работать, как самостоятельно, так и в комбинации с разного рода нагревательными приборами | 1921 |
|
SU10A1 |
| УСТРОЙСТВО ДЛЯ ПЕРЕРАБОТКИ СЫРОГО ТОРФА НА ТОРФЯНЫЕ КИРПИЧИ | 1918 |
|
SU722A1 |
| ЛОБАНОВА Н.В | |||
| и др | |||
| Клинические, иммуногистохимические и молекулярно-генетические факторы прогноза у | |||
Авторы
Даты
2024-07-17—Публикация
2023-09-29—Подача