Список последовательностей
Настоящая заявка содержит Перечень последовательностей, который представлен в электронном виде в формате ASCII и включен в настоящий документ путем отсылки во всей своей полноте. Копия ASCII, созданная 12 апреля 2022 года, называется «G4590-15000PCT_SeqListing_20220412» и имеет размер 111 килобайт.
Перекрестные ссылки на имеющие отношение заявки
Настоящая заявка претендует на все преимущества и приоритет предварительной заявки на патент США № 63/173,752, зарегистрированной 12 апреля 2021 г., и предварительной заявки на патент США № 63/264,737 зарегистрированной 1 декабря 2021 г., раскрытие которых включено в настоящий документ путем отсылки во всей своей полноте.
Область техники, к которой относится изобретение
Настоящее раскрытие в целом относится к области лечения и/или профилактики коронавирусной инфекции. В особенности, настоящее раскрытие относится к вакцинам на основе информационной РНК (мРНК) против широкого спектра вариантов коронавируса (CoV).
Уровень техники
В 1796 году Эдвард Дженнер создал первую в мире вакцину (коровьей оспы) для защиты от оспы и успешно спас миллионы людей. С тех пор вакцинация признана лучшим способом защиты от патогенов. С момента вспышки тяжелого острого респираторного синдрома коронавирус-2 (SARS-CoV-2) в декабре 2019 года, вызвавшего индуцированное коронавирусом заболевание 2019 (COVID-19), вирус распространился по всему миру и вызвал более 200 миллионов случаев инфицирования и 4 миллионов смертей за 20 месяцев. Эта пандемия стала серьезной угрозой для общественного здравоохранения.
Много усилий было направлено на разработку эффективных средств и вакцин для борьбы с этой пандемией. Трехмерный шиповидный белок (S) на поверхности вируса был ключевым иммуногеном и мишенью для разработки профилактических вакцин и терапевтических антител. По состоянию на декабрь 2020 года Управление по контролю качества пищевых продуктов и лекарственных средств США разрешило применять кандидатные мРНК-вакцины Pfizer/BioNTech и Moderna, а также антитела Regeneron в экстренных случаях; однако несколько других вакцин-кандидатов и человеческие антитела проходят клинические испытания, и некоторые из них, включая вакцины Oxford/Astrazeneca и J&J, близки к получению одобрения. Из различных вакцин, разработанных для контроля распространения SARS-CoV-2 и его вариантов, мРНК-вакцины, разработанные Moderna и BioNTech/Pfizer, представляют собой крупный прорыв благодаря их скорости и удобству. Эти вакцины были стабилизированы с помощью новой технологии мРНК и композиции липидных наночастиц (LNP) для доставки и трансляции в шиповидный белок (S) in vivo для индукции иммунного ответа (Ewen Callaway. COVID vaccine excitement builds as Moderna reports third positive result. Nature. 587 (7834):337-338 (2020); Polack P. et al. Safety and Efficacy of the BNT162b2 mRNA Covid-19 Vaccine. N Engl J Med. 383 (27):2603-2615 (2020)).
Однако, поскольку существует ряд вариантов, которые широко циркулируют в мире, и более инфекционные варианты, такие как варианты бета, дельта и омикрон, могут иметь повышенную способность повторно заражать людей, которые были вакцинированы ранее и/или выздоровели от инфекции более ранними версиями коронавируса, инфекции новыми вариантами SARS-CoV-2 могут продолжать происходить или потенциально увеличиваться по частоте. S-белок этого РНК-вируса сильно гликозилирован и часто мутирует с более чем 9 миллионами последовательностей и более чем 1000 сайтов мутации в его 1273 аминокислотных последовательностях, о чем сообщает GISAID (www.gisaid.org), включая легко передающиеся дельта и омикрон варианты, представляющие серьезную проблему в разработке широко эффективных антител и вакцин.
Соответственно, существует насущная потребность в более качественных вакцинах, а также в более качественных продуктах и способах для профилактики и лечения коронавирусных инфекций.
Раскрытие изобретения
Настоящее изобретение обеспечивает новую мРНК-вакцину против коронавируса, способы ее получения и применения. Новая вакцина разработана на основе технологии мРНК для удаления гликановых экранов шиповидного белка коронавируса (например, SARS-CoV-2), чтобы лучше обнажить консервативные области шиповидного белка. В мРНК-вакцине с шиповидным белком коронавируса делеция сайтов гликозилирования в рецептор-связывающем домене (RBD) или домене субъединицы 2 (S2) приводит к экспонированию высококонсервативных эпитопов и выработке антител и CD8 Т-клеточного ответа с более широкой защитой от альфа-, бета-, гамма, дельта, омикрон и различных вариантов по сравнению с немодифицированной мРНК. Представленная в настоящем документе мРНК-вакцина эффективна для индуцирования защитного иммунитета против SARS-CoV-2 и его вариантов (например, альфа, бета, гамма, дельта, омикрон). При применении по отдельности или в комбинации в качестве иммуногенной композиции или вакцины, мРНК-вакцина по настоящему изобретению может защищать людей от инфекции и/или уменьшать симптомы при инфицировании.
В одном аспекте настоящее изобретение обеспечивает, по меньшей мере, один иммуногенный пептид, включающий аминокислотную последовательность, выбранную из группы, состоящей из: TESIVRFPNITNL (SEQ ID NO:41), NITNLCPFGEVFNATR (SEQ ID NO:42), LYNSASFSTFK (SEQ ID NO:43), LDSKVGGNYN (SEQ ID NO:44), KSNLKPFERDIST (SEQ ID NO:45), KPFERDISTEIYQAG (SEQ ID NO:46), GPKKSTNLVKNKC (SEQ ID NO:47), NCDVVIGIVNNTVY (SEQ ID NO:48), PELDSFKEELDKYFK[N]HTS (SEQ ID NO:49), VNIQKEIDRLNEVA (SEQ ID NO:50), NLNESLIDLQ (SEQ ID NO:51) и LGKYEQYIKWP (SEQ ID NO:52) или аминокислотную последовательность, имеющую, по меньшей мере, примерно 99%, 98%, 97%, 96%, 95% или 90% идентичности любой из последовательностей SEQ ID NOs: 41-52.
В некоторых воплощениях, иммуногенный пептид включает, по меньшей мере, аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 41-43 и 45-51. В некоторых воплощениях иммуногенный пептид включает, по меньшей мере, одну, две, три, четыре, пять, шесть, семь, восемь, девять, десять, одиннадцать или двенадцать аминокислот SEQ ID NO: 41-52. В некоторых воплощениях иммуногенный пептид включает, по меньшей мере, одну, две, три, четыре, пять, шесть, семь, восемь, девять, десять аминокислот SEQ ID NO:SEQ ID NO: 41-43 и 45-51.
В одном аспекте настоящее изобретение относится к молекуле модифицированной нуклеиновой кислоты, кодирующей модифицированный шиповидный белок, включающий одну или несколько аминокислотных замен в сиквонах N-связанного гликозилирования (N-X-S/T), где X представляет собой любой аминокислотный остаток, кроме пролина, и S/Т обозначает остаток серина или треонина.
В некоторых воплощениях, модифицированный шиповидный белок, описанный в настоящем документе, включает замену аспарагина (N) на глутамин (Q) в сиквонах N-связанного гликозилирования (N-X-S/T) для удаления сиквонов N-связанного гликана.
В некоторых воплощениях, модифицированный шиповидный белок, описанный в настоящем документе, включает замену одной или нескольких аминокислот в сиквонах N-связанного гликозилирования (N-X-S/T) для удаления сиквонов N-связанного гликана.
В некоторых воплощениях, модифицированный шиповидный белок, описанный в настоящем документе, включает одну или несколько аминокислотных замен S/T в O-связанных сайтах гликозилирования для устранения O-связанных сайтов гликозилирования. Один из примеров представляет собой замену S/T на аланин (А).
В одном воплощении, модифицированная молекула нуклеиновой кислоты представляет собой мРНК или двухцепочечную или одноцепочечную ДНК.
В одном воплощении, модифицированный шиповидный белок получают из шиповидного белка SARS-CoV-2. Шиповидный белок SARS-CoV-2, описанный в настоящем документе, включает аминокислотную последовательность SEQ ID NO: 2, 16, 18 или 20 или аминокислотную последовательность, имеющую, по меньшей мере, примерно 99%, 98%, 97%, 96%, 95%, 90%, 85% или 80% идентичности аминокислотной последовательности SEQ ID NO: 2, 16, 18 или 20.
В некоторых воплощениях, молекула нуклеиновой кислоты, кодирующая аминокислотную последовательность SEQ ID NO: 2, 16, 18 или 20, представляет собой мРНК, включающую нуклеотидную последовательность SEQ ID NO: 1, 15, 17 или 19, соответственно, или нуклеотидную последовательность, имеющую, по меньшей мере, приблизительно 99%, 98%, 97%, 96%, 95%, 90%, 85% или 80% идентичности нуклеотидной последовательности SEQ ID NO: 1, 15, 17 или 19, соответственно.
В некоторых воплощениях, модифицированный шиповидный белок, описанный в настоящем документе, включает аминокислотную последовательность SEQ ID NO: 4, 22, 24 или 26, в которой модифицированный шиповидный белок включает домен связывания рецептора (RBD), в котором отсутствуют сайты гликозилирования. Молекула модифицированной нуклеиновой кислоты, кодирующая аминокислотную последовательность SEQ ID NO: 4, 22, 24 или 26, включает нуклеотидную последовательность SEQ ID NO: 3, 21, 23 или 25, соответственно, или нуклеотидную последовательность, имеющую, по меньшей мере, примерно 99%, 98%, 97%, 96%, 95%, 90%, 85% или 80% идентичности нуклеотидной последовательности SEQ ID NO: 3, 21, 23 или 25, соответственно.
В некоторых воплощениях, модифицированный шиповидный белок, описанный в настоящем документе, включает аминокислотную последовательность SEQ ID NO: 6, 28, 30 или 32, в которой модифицированный шиповидный белок включает субъединицу S2, лишенную сайтов гликозилирования. Молекула модифицированной нуклеиновой кислоты, кодирующая аминокислотную последовательность SEQ ID NO: 6, 28, 30 или 32, включает нуклеотидную последовательность SEQ ID NO: 5, 27, 29 или 31, соответственно, или нуклеотидную последовательность, имеющую, по меньшей мере, примерно 99%, 98%, 97%, 96%, 95%, 90%, 85% или 80% идентичности нуклеотидной последовательности SEQ ID NO: 527, 29 или 31, соответственно.
В некоторых воплощениях, модифицированный шиповидный белок, описанный в настоящем документе, включает аминокислотную последовательность SEQ ID NO: 8 или 34, в которой модифицированный шиповидный белок включает субъединицу S2, которая состоит из одного сайта гликозилирования. В некоторых воплощениях единственный сайт гликозилирования находится в положении N1194. В некоторых воплощениях молекула модифицированной нуклеиновой кислоты, кодирующая аминокислотную последовательность SEQ ID NO: 8 или 34, включает нуклеотидную последовательность SEQ ID NO: 7 или 33, соответственно, или нуклеотидную последовательность, имеющую, по меньшей мере, примерно 99%, 98%, 97%, 96%, 95%, 90%, 85% или 80% идентичность нуклеотидной последовательности SEQ ID NO: 7 или 33, соответственно.
В некоторых воплощениях, модифицированный шиповидный белок, описанный в настоящем документе, включает аминокислотную последовательность SEQ ID NO: 10 или 36, в которой модифицированный шиповидный белок включает домен связывания рецептора (RBD), в котором отсутствуют сайты гликозилирования, и аминокислотную замену N801 на Q801. Молекула модифицированной нуклеиновой кислоты, кодирующая аминокислотную последовательность SEQ ID NO: 10 или 36, включает нуклеотидную последовательность SEQ ID NO: 9 или 35, соответственно, или нуклеотидную последовательность, имеющую, по меньшей мере, примерно 99%, 98%, 97%, 96%, 95%, 90%, 85% или 80% идентичности нуклеотидной последовательности SEQ ID NO: 9 или 35 соответственно.
В некоторых воплощениях, модифицированный шиповидный белок, описанный в настоящем документе, включает аминокислотную последовательность SEQ ID NO: 12 или 38, в которой модифицированный шиповидный белок включает домен связывания рецептора (RBD), в котором отсутствуют сайты гликозилирования, и аминокислотную замену N1194 на Q1194. Молекула модифицированной нуклеиновой кислоты, кодирующая аминокислотную последовательность SEQ ID NO: 12 или 38, включает нуклеотидную последовательность SEQ ID NO: 11 или 37, соответственно, или нуклеотидную последовательность, имеющую, по меньшей мере, примерно 99%, 98%, 97%, 96%, 95%, 90%, 85% или 80% идентичности нуклеотидной последовательности SEQ ID NO: 11 или 37, соответственно.
В некоторых воплощениях, модифицированный шиповидный белок, описанный в настоящем документе, включает аминокислотную последовательность SEQ ID NO: 14 или 40, в которой модифицированный шиповидный белок включает модифицированный домен связывания рецептора (RBD), лишенный сайтов гликозилирования, и аминокислотные замены N122 на Q122, N165 на Q165 и N234 на Q234. Молекула модифицированной нуклеиновой кислоты, кодирующая аминокислотную последовательность SEQ ID NO: 14 или 40, включает нуклеотидную последовательность SEQ ID NO: 13 или 39, соответственно, или нуклеотидную последовательность, имеющую, по меньшей мере, примерно 99%, 98%, 97%, 96%, 95%, 90%, 85% или 80% идентичности нуклеотидной последовательности SEQ ID NO: 13 или 39 соответственно.
В некоторых воплощениях, описанный в настоящем документе модифицированный шиповидный белок включает субъединицу S1, в которой отсутствуют сайты гликозилирования.
В некоторых воплощениях, описанный в настоящем документе модифицированный шиповидный белок включает как субъединицу S1, так и S2, в которых отсутствуют сайты гликозилирования.
Настоящее изобретение относится к мРНК-вакцине шиповидного белка коронавируса с делецией сайтов гликозилирования в рецептор-связывающем домене (RBD) или домене субъединицы 2 (S2) для экспонирования высококонсервативных эпитопов и индукции антител и Т-клеточного ответа CD8 с более широкой защитой от альфа, бета, гамма, дельта, омикрон и различных вариантов по сравнению с немодифицированной мРНК.
В некоторых воплощениях, вакцина против коронавируса включает мРНК шиповидного белка коронавируса с одной или несколькими мутациями сайтов гликозилирования в RBD или S2 или других доменах с одной или несколькими заменами N на Q или S/T на A, или их комбинацией. В другом воплощении мутация сайтов N-гликозилирования заключается в изменении предполагаемой последовательности N-X-S/T на Q-X-S/T и/или изменении S/T на A в сайте О-гликозилирования.
В некоторых воплощениях, мРНК, описанные в настоящем документе, имеющие сайты гликозилирования с заменой N на Q, включают S-(deg-RBD) (белок S, в котором все 2 N-сайты гликозилирования в RBD мутированы с N на Q, а 2 O-сайты гликозилирования мутированы с S/T на A), S-(deg-S2) (белок S со всеми 9 сайтами гликозилирования в S2, мутированными с N на Q), S-(deg-S2-1194) (белок S с 8 сайтами гликозилирования в S2, мутировавшими с N на Q, кроме сайта гликозилирования 1194), S-(deg-RBD-801) (белок S, в котором все 2 N-сайты гликозилирования в RBD мутированы с N на Q, а 2 O-сайты гликозилирования мутированы с S/T на A, а сайт гликозилирования 801 мутирован с N на Q), S-(deg-RBD-1194) (белок S со всеми 2 N-сайтами гликозилирования в RBD, мутированными с N на Q, и 2 сайта О-гликозилирования, мутированными с S/T на A, и сайт гликозилирования 1194, мутированный с N на Q), и S-(deg-RBD-122-165-234) (S-белок со всеми 2 N-сайтами гликозилирования в RBD, мутированными с N на Q, и 2 сайта О-гликозилированиями, мутированными с S/T на A, и сайтами гликозилирования 122, 165 и 234, мутированными от N до Q).
В одном воплощении, иммунизация типовой коронавирусной вакциной по настоящему изобретению, как описана в настоящем документе, приводит к накоплению неправильно свернутого белка S в эндоплазматическом ретикулуме. В одном из случаев иммунизация типовой коронавирусной вакциной по настоящему изобретению, как описана в настоящем документе, вызывает активацию BiP/GRP78, XBP1 и p-eIF2α, вызывающую апоптоз клеток и CD8+ T-клеточный ответ. В одном из случаев иммунизация коронавирусной вакциной по настоящему изобретению, как описано в настоящем документе, может повышать экспрессию главного комплекса гистосовместимости класса I (MHC I).
В некоторых воплощениях, типовые CoV, описанные в настоящем документе, включают, но не ограничиваются ими, SARS-CoV, MERS-CoV и SARS-CoV-2. SARS-CoV2, бета-SARS-CoV2, гамма-SARS-CoV2, дельта-SARS-CoV2 и омикрон-SARS-CoV2 и их варианты.
В некоторых воплощениях, настоящее изобретение относится к линейной ДНК, включающей промотор, 5'-нетранслируемую область, 3'-нетранслируемую область, экспрессионную плазмиду с S-2P или без него и сигнальную последовательность поли(А)-хвоста, в которой предполагаемая последовательность N-X-S/T заменена на Q-X-S/T, а сайт О-гликозилирования был заменен с S/T на A на экспрессионной плазмиде.
В некоторых воплощениях, экспрессионная плазмида S-2P включает ген S вируса SARS-CoV-2, кодирующий S в состоянии до слияния с заменами пролина K968 и V969.
В некоторых воплощениях, настоящее изобретение обеспечивает мРНК, полученную путем трансляции in vitro из вышеупомянутой ДНК.
В другом аспекте, настоящее изобретение обеспечивает вектор, включающий модифицированную молекулу нуклеиновой кислоты, описанную выше.
В другом аспекте, настоящее изобретение относится к клетке-хозяину, включающей модифицированную молекулу нуклеиновой кислоты, описанную выше.
В другом аспекте, настоящее изобретение обеспечивает модифицированный шиповидный белок, описанный выше.
В другом аспекте, в настоящем изобретении предложен способ доставки мРНК для продукции белка in vivo, включающий: введение субъекту композиции, включающей мРНК по изобретению, которая кодирует белок, в котором мРНК инкапсулирована в липидную наночастицу, и в котором введение композиции приводит к экспрессии белка, кодируемого мРНК.
В некоторых воплощениях, описанная в настоящем документе мРНК может быть применена в качестве вакцины либо отдельно, либо в комбинации с другими вакцинами. Соответственно, настоящее изобретение обеспечивает комбинированную вакцину, включающую мРНК-вакцину по настоящему изобретению и одну или несколько дополнительных вакцин. Дополнительная вакцина выбрана из одной или нескольких вакцин против COVID-19, вакцины против гриппа, адвеновирусной вакцины, вакцины против сибирской язвы, вакцины против холеры, вакцины против дифтерии, вакцины против гепатита А или В, вакцины против HPV, вакцины против кори, вакцины против эпидемического паротита, вакцины против оспы, ротавирусной вакцины, противотуберкулезной вакцины, пневмококковой вакцины и вакцины против Haemophilus influenzae типа b и любой их комбинации.
В другом аспекте, настоящее изобретение обеспечивает наночастицу на основе гуанидина, применяемую в качестве носителя для доставки модифицированной молекулы нуклеиновой кислоты по любому из пп. 1-25 субъекту. В одном воплощении наночастица представляет собой липосому или полимерсому.
В другом аспекте, настоящее изобретение обеспечивает нанокластер мРНК, включающий мРНК-вакцину, описанную в настоящем документе, в составе липидных наночастиц. В одном воплощении липидная наночастица представляет собой биоразлагаемую липидную наночастицу.
В некоторых воплощениях, липидная наночастица, как описана в настоящем документе, представляет собой полимеры на основе гуанидина. В одном воплощении настоящее изобретение относится к нанокластеру мРНК, включающему липидные наночастицы, инкапсулированные с мРНК-вакциной, описанной в настоящем документе, при этом липидная наночастица включает полимерные звенья на основе гуанидина, где гуанидиновые, а также цвиттерионные группы полимера присоединяются к липидному хвосту полимера, и где полимеры на основе гуанидина прикрепляются к мРНК, тем самым образуя солевые мостики между гуанидиновыми группами и фосфатами в мРНК. Примеры полимеров на основе гуанидина включают, но не ограничиваются ими, P1, P2, P3, Pb и Pz, как показано ниже.
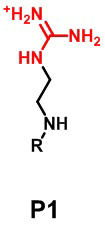 ,
, 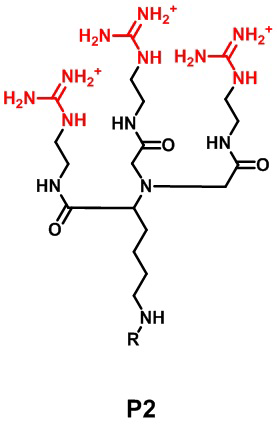 ,
,  ,
,  и
и 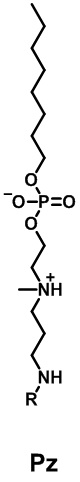 , в которых R представляет собой
, в которых R представляет собой 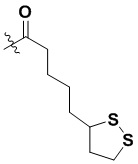 .
.
В некоторых воплощениях, полимер на основе гуанидина образует сополимер, такой как сополимер P1/P3, сополимер P2/P3, сополимер P1/Pb, сополимер P2/Pb, сополимер P1/Pz и сополимер P2/Pz.
В некоторых воплощениях, наночастицы на основе гуанидина и цвиттерионных липидов включают смесь P1 и/или P2 и Pz, ,
, ,
,  , в которых R представляет собой
, в которых R представляет собой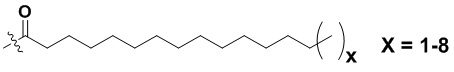 или
или  .
.
В некоторых воплощениях, нанокластер мРНК, описанный в настоящем документе, имеет отношение наночастица/мРНК (N/P), равное примерно 1, примерно 2, примерно 3, примерно 4, примерно 5, примерно 6, примерно 7, примерно 8, примерно 9, примерно 10, примерно 15, примерно 20, примерно 30, примерно 40, примерно 50 или примерно 100.
В некоторых воплощениях, нанокластер мРНК, описанный в настоящем документе, имеет соотношение наночастиц/мРНК (N/P), равное примерно 10 или примерно 20.
В некоторых воплощениях, настоящее изобретение относится к композиции наночастиц/нанокластеров, включающей наночастицы, присоединенные к вакцине против коронавируса согласно настоящему изобретению. В одном воплощении наночастица представляет собой липидную наночастицу, полимерную наночастицу, неорганическую наночастицу, такую как наночастица золота, липосому, иммуностимулирующий комплекс, вирусоподобную частицу или самособирающийся белок. В другом воплощении наночастица представляет собой липидную наночастицу (LNP).
В некоторых воплощениях, настоящее изобретение относится к вакцинной композиции, включающей мРНК-вакцину, нанокластер/нанокластер мРНК или композицию наночастиц, как описано в настоящем документе.
В некоторых воплощениях, настоящее изобретение относится к антителам и CD8+ Т-клеткам, индуцированным вакциной, описанной в настоящем документе, которые обладают более широкой защитой от альфа-, бета-, гамма-, дельта- и омикронных вариантов.
В некоторых воплощениях, в настоящем изобретении предложен способ иммунизации субъекта, включающий введение композиции вакцины, описанной в настоящем документе. В настоящем изобретении также предложен способ профилактики или лечения коронавирусной инфекции, включающий введение эффективного количества мРНК-вакцины, нанокластера мРНК или наночастицы мРНК или вакцинной композиции, как описаны в настоящем документе, субъекту, инфицированному или подверженному риску заражения коронавирусом. В одном воплощении мРНК-вакцина, нанокластер мРНК или наночастица мРНК или вакцинная композиция, как описаны в настоящем документе, могут быть применены в способе усиления адаптивного иммунного ответа.
В некоторых воплощениях, РНК-вакцину, нанокластер мРНК или наночастицу мРНК или вакцинную композицию, как описаны в настоящем документе, вводят в виде начальной дозы и двух, трех или четырех бустерных доз. В некоторых воплощениях мРНК-вакцину, нанокластер мРНК или наночастицу мРНК или вакцинную композицию вводят в начальной дозе и, по меньшей мере, в одной бустерной дозе в течение примерно одного месяца, примерно двух месяцев, примерно трех месяцев, примерно четырех месяцев, примерно пяти месяцев или примерно через шесть месяцев после первоначальной дозы. В некоторых воплощениях обеспечиваемую композицию вводят во второй бустерной дозе приблизительно через шесть месяцев, приблизительно через семь месяцев, приблизительно через восемь месяцев, приблизительно через девять месяцев, приблизительно через десять месяцев, приблизительно через одиннадцать месяцев или приблизительно через год после введения начальной дозы.
В некоторых воплощениях, мРНК-вакцину, нанокластер мРНК или наночастицу мРНК или вакцинную композицию вводят в виде одной или нескольких доз. В одном воплощении доза может включать или исключать от 5 мкг до 50 мкг мРНК. В некоторых воплощениях доза составляет примерно 5 мкг, 10 мкг, 15 мкг, 20 мкг, 25 мкг, 30 мкг, 35 мкг, 40 мкг, 45 мкг или 50 мкг.
В некоторых воплощениях, мРНК-вакцину, нанокластер мРНК или наночастицу мРНК или вакцинную композицию вводят внутривенно, внутримышечно, внутрикожно или подкожно, или путем инфузии или назального спрея.
В некоторых воплощениях, в настоящем изобретении предложен способ получения вакцин и антител с широким спектром защиты против SARS-CoV-2. В одном воплощении способ включает создание вакцины с применением РНК или ДНК нативного S-белка или S-белка, полученного с помощью гликоинженерии, тогда как белок, экспрессируемый в антиген-презентирующих клетках, включая свернутые или развернутые формы, обрабатывается и презентируется Т-клеткам.
Эти и другие аспекты станут очевидными из следующего описания предпочтительного воплощения, взятого в сочетании со следующими чертежами, хотя возможны изменения и модификации, не отступающие от сущности и объема новых концепций раскрытия.
Краткое описание чертежей
Следующие чертежи составляют часть настоящего описания и включены для дополнительной демонстрации определенных аспектов настоящего раскрытия, изобретения которого можно лучше понять, обратившись к одному или нескольким из этих чертежей в сочетании с подробным описанием конкретных воплощений, представленных в настоящем документе.
Фиг. 1. Консервативные эпитопы вариантов S-белка, 10 из которых экранированы гликанами.
Фиг. 2. Идентификация N- и O-сайтов гликозилирования и мутаций в вариантах. Все 24 сайта гликозилирования высококонсервативны среди 6 миллионов последовательностей белков S.
Фиг. 3. Анализ экспрессии белка S после трансфекции мРНК через 48 часов с помощью вестерн-блоттинга. Фильтр исследовали с помощью моноклональных антител против S и против β-актина.
Фиг. 4A-4F. Гуморальный иммунный ответ у мышей BALB/c был показан как титр в сыворотке крови конечной точки IgG, специфичного к белку анти-S WT (A), S2 (B), RBD (C), дегликозилированного S (D), дегликозилированного S2 (E) и дегликозилированного белка RBD (F), полученный с помощью ELISA. Среднее ± SD для пяти независимых экспериментов. *P < 0,001.
Фиг. 5А-5D. Гликозилирование регулировало специфичность индуцированных антител и влияло на степень защиты мРНК-вакцины. Конечная точка определенного с помощью ELISA титра антител IgG, специфичных к альфа (А), бета (В), гамма (С) и дельта (D) S-белку. Среднее ± SD для пяти независимых экспериментов. *P < 0,001, **P < 0,05.
Фиг. 6А-6F. Кривые нейтрализации вариантов псевдовируса показаны в WT (A), альфа (B), бета (C), гамма (D), дельта (E) и омикрон (F). Среднее ± SD для пяти независимых экспериментов. *P < 0,001, **P < 0,05.
Фиг. 7A-7E. Гликозилирование влияло на ответ CD8+ T-клеток. Спленоциты, выделенные из иммунизированных мышей, инкубировали с пулами полноразмерных пептидов WT S (A), RBD (B) и S2 (C), затем измеряли GrzB-секретирующие клетки с помощью Elispot. CD4+ (D) и CD8+ (E) Т-клетки выделяли и инкубировали с дендритными клетками костного мозга и пулом полноразмерных пептидов WTS для измерения IFNγ-секретирующих Т-клеток с помощью проточной цитометрии. (A-E) Среднее ± SD для пяти независимых экспериментов. *P < 0,001.
Фиг. 8A-8F. Гликозилирование влияет на продукцию цитокинов. После инкубации спленоцитов, выделенных из мышей, иммунизированных мРНК-вакциной, с пулом полноразмерных пептидов WTS S, определяли (A) IFNγ, (B) IL-2, (C) IL-4, (D) IL-6, (E) IL-12 и (F) IL-13. (A-F) Среднее ± SD для пяти независимых экспериментов. *P < 0,001, **P < 0,05.
Фиг. 9. Делеция сайтов гликозилирования в мРНК с образованием дегликозилированного S-белка и реакция развернутого белка. Анализ экспрессии дегликозилированного белка S с помощью клеток HEK293T, трансфицированных плазмидами и обработкой MG132, с помощью вестерн-блоттинга. Фильтр анализировали с помощью моноклональных антител против S и против GAPDH.
Фиг. 10. In vitro транслированные дегликозилированные варианты S в разное время инкубации, как показано на фигуре, контролировали с помощью ELISA. Среднее ± SD для трех независимых экспериментов. *P < 0,001.
Фиг. 11A-11C. После того как клетки HEK293 трансфицировали мРНК-вакциной через 48 часов, выделяли плазматическую мембрану (A), цитозоль (без ER) (B) и ER (C) для анализа количества белка S с помощью вестерн-блоттинга. Фильтр анализировали с помощью моноклональных антител против S, против Na/K-АТФазы, против SERCA2 и против GAPDH.
Фиг. 12. Анализ маркерных белков UPR BiP/GRP78, XBP1 и p-eIF2α с помощью вестерн-блоттинга после трансфекции клеток HEK293 мРНК-вакциной дегликозилированных вариантов белка S через 48 часов. Фильтр анализировали с помощью моноклональных антител анти-BiP, анти-XBP1, анти-p-eIF2α и анти-β-актин.
Фиг. 13. Для анализа апоптотических клеток с помощью анализа APO-BrdU TUNEL после того, как клетки HEK293 были трансфицированы мРНК-вакциной в разное время, как показано на рисунке. Среднее ± SD для трех независимых экспериментов. *P < 0,001.
Фиг. 14. Анализ экспрессии MHC I с помощью проточной цитометрии DC после инкубации с вариантами мРНК-вакцин. Среднее ± SD для трех независимых экспериментов. *P < 0,001.
Фиг. 15A-15G. Схематическое изображение шиповидного белка SARS-CoV-2 и конструкции вакцины: WT (A); S-(deg-RBD) (B); S-(deg-S2) (C); S-(deg-S2-1194) (D); S-(deg-RBD-801) (E); S-(deg-RBD-1194) (F); S-(deg-RBD-122-165-234) (G). NTD, N-концевой домен (14-305 остатков). RBD, рецептор-связывающий домен (319-541 остатков). FP, слитый пептид (788-806 остатков). HR1, последовательность 1 гептапептидного повтора (912-984 остатка). HR2, последовательность 2 гептапептидного повтора (1163-1213 остатков). TM, трансмембранный домен (1213-1237 остатков). CT, цитоплазматический домен (1237-1273 остатка). субъединица S2 (686-1273 остатка). 2P, (K986P и V987P). Ψ - сайт N-гликозилирования; ϕ - сайт O-гликозилирования.
Фиг. 16. Гликозилирование S2 регулировало секрецию растворимого шиповидного белка SARS-CoV-2 перед слиянием. После того как клетки HEK293 трансфицировали мРНК-вакциной, которая кодировала растворимую версию варианта S до слияния, местоположение S определяли вестерн-блоттингом. Фильтр анализировали с помощью моноклональных антител против S и против GAPDH.
Фиг. 17. мРНК-вакцина влияла на экспрессию MHC II на DC. Анализ экспрессии MHC II с помощью проточной цитометрии DC после инкубации с вариантами мРНК-вакцин. Среднее ± SD для трех независимых экспериментов. *P < 0,001.
Фиг. 18A-18C. Характеристика иммунного ответа на мРНК-вакцину с делецией специфического сайта гликозилирования в S. Гуморальный иммунный ответ у мышей BALB/c был продемонстрирован как белок-специфический титр IgG из сыворотки против S WT (A), RBD (B) и дегликозилированного RBD (C), проанализированный с помощью ELISA.
Фиг. 19A-19E. Характеристика иммунного ответа на мРНК-вакцину с делецией специфического сайта гликозилирования в S. Кривые нейтрализации вариантов псевдовируса показаны с WT (A), альфа (B), бета (C), гамма (D) и дельта (E).
Фиг. 20A-20B. Характеристика иммунного ответа на мРНК-вакцину с делецией специфического сайта гликозилирования в S. (A) После инкубации спленоцитов, выделенных от иммунизированных мышей, с пулами полноразмерных пептидов WTS, GrzB-секретирующие клетки измеряли с помощью Elispot. (B) CD8+ Т-клетки от иммунизированных мышей выделяли и инкубировали с DC костного мозга и пулом полноразмерных пептидов WTS для измерения IFNγ-секретирующих Т-клеток с помощью проточной цитометрии. (A-J) Среднее ± SD для пяти независимых экспериментов. *P < 0,001.
Фиг. 21A и 21B. Уровень экспрессии белка с делецией специфического сайта гликозилирования в S. (A) Анализ экспрессии различных белков S с помощью клеток HEK293T, трансфицированных плазмидами, и обработки MG132 с помощью вестерн-блоттинга. (B) Анализ экспрессии белка S в клетках HEK293T после трансфекции мРНК-LNP через 48 часов с помощью вестерн-блоттинга. Фильтр анализировали с помощью моноклональных антител против S и против GAPDH.
Фиг. 22A-22C. Характеристика иммунного ответа на мРНК-вакцину с делецией специфического сайта гликозилирования в S. После инкубации спленоцитов, выделенных от иммунизированных мышей, с пулами пептидов RBD (A) и S2 (B), GrzB-секретирующие клетки измеряли с помощью Elispot. (C) CD4+ T-клетки выделяли из иммунизированных мышей и инкубировали с DC костного мозга и пулом полноразмерных пептидов WTS для измерения IFNγ-секретирующих T-клеток с помощью проточной цитометрии. (A-C) Среднее ± SD для пяти независимых экспериментов. *P < 0,001.
Фиг. 23. Пропагатор P1, содержащий гуанидиновые группы, и мультивалентный дисплейный пропагатор P2 облегчают сцепление мРНК с полимерами за счет образования прочных солевых мостиков между гуанидинами и фосфатами в мРНК.
Фиг. 24. Разработанная структура инициаторов (I0), пропагаторов (P1, P2, Pb, P3, Pz) и процесс полимерной реакции.
Фиг. 25A и 25B. (A) Анализ электрофореза сополимеров с мРНК GFP в агарозном геле при соотношении N/P = 10. (B) Размер частиц комплексов мРНК, визуализированный с помощью TEM.
Фиг. 26A-26C. Флуоресцентное изображение GFP-экспрессии мРНК GFP, трансфицированной поли(дисульфидами) в клетках HEK293T. (A) мРНК GFP в комплексе с P1, P3, P1/P3 и PEI в соотношении N/P = 10 (B) мРНК GFP в комплексе с P1/P3, P2/P3, P1/Pb и P2/Pb P1/Pz в соотношении N/P = 10 (C) мРНК GFP в комплексе с различным соотношением N/P.
Фиг. 27. Анализ электрофореза в агарозном геле комплексов мРНК шиповидного белка-полимер при различных соотношениях N/P.
Фиг. 28. Хемилюминесцентная визуализация экспрессии шиповидного белка, опосредованной комплексами мРНК шиповидного белка-полимер в клетках HEK293T.
Фиг. 29. Анализ клеточной жизнеспособности клеток HEK293T после обработки различными количествами комплексов полиГу/мРНК шиповидного белка. Соотношение варьировали от 0,01 до 1. Планка погрешности представляет собой стандартную ошибку (среднее ± S.D., n=3).
Осуществление изобретения
Термины, применяемые в этом описании, обычно имеют свои обычные значения в данной области техники, в контексте изобретения и в конкретном контексте, в котором применяют каждый термин. Определенные термины, которые применяют для описания изобретения, обсуждаются ниже или в другом месте описания, чтобы обеспечить практикующему врачу дополнительные указания относительно описания изобретения. Для удобства некоторые термины могут быть выделены, например, курсивом и/или кавычками. Применение выделения не влияет на объем и значение термина; объем и значение термина остаются теми же в одном и том же контексте, независимо от того, выделен он или нет. Следует понимать, что одно и то же можно сказать более чем одним способом. Следовательно, для любого одного или нескольких обсуждаемых в настоящем документе терминов могут применяться альтернативные формулировки и синонимы, и не следует придавать особого значения тому, разрабатывается или обсуждается ли термин в данном документе. Обеспечены синонимы к некоторым терминам. Упоминание одного или нескольких синонимов не исключает применения других синонимов. Применение примеров в любом месте данного описания, включая примеры любых обсуждаемых в настоящем документе терминов, носит исключительно иллюстративный характер и никоим образом не ограничивает объем и значение изобретения или любого приведенного в качестве примера термина. Точно так же изобретение не ограничено различными воплощениями, приведенными в данном описании.
Следует отметить, что термины в единственном числе, применяемые в данном описании и прилагаемой формуле изобретения, охватывают и множественное число, если содержание явно не требует иного.
Как применены в настоящем документе, термины «шиповидный белок» и «шиповидный гликопротеин» и «шиповидный белок коронавируса» применяют как взаимозаменяемые.
Как применены в настоящем документе, термины «дикого типа (нативный) шиповидный белок коронавируса», «дикого типа (нативный) шиповидный гликопротеин коронавируса», «дикого типа (нативный)шиповидный гликопротеин» и «дикого типа (нативный)шиповидный белок» применяют взаимозаменяемо.
Как применены в настоящем документе, термины «лечить», «лечение» и «проведение лечения» относятся к подходу для получения полезных или желаемых результатов, например, клинических результатов. Для целей настоящего описания благоприятные или желаемые результаты могут включать ингибирование или подавление возникновения или прогрессирования инфекции или заболевания; облегчение или уменьшение развития симптомов инфекции или заболевания; или их комбинация.
Как применены в настоящем документе, термины «предупреждение» и «превенция» применяют взаимозаменяемо с термином «профилактика», и они могут означать полное предотвращение инфекции или предотвращение развития симптомов этой инфекции; задержку начала инфекции или ее симптомов; или уменьшение тяжести впоследствии развившейся инфекции или ее симптомов.
Как применено в настоящем документе, «эффективное количество» относится к количеству иммуногена, достаточному для индукции иммунного ответа, который уменьшает, по меньшей мере, один симптом патогенной инфекции. Эффективную дозу или эффективное количество можно определить, например, путем измерения количества нейтрализующих секреторных и/или сывороточных антител, например, путем нейтрализации бляшек, фиксации комплемента, твердофазного иммуноферментного анализа (ELISA) или анализа микронейтрализации.
Как применен в настоящем документе, термин «вакцина» относится к иммуногенному средству (с адъювантом или без него), такому как иммуноген, полученный из коронавируса, который применяют для индукции иммунного ответа против коронавируса, который обеспечивает защитный иммунитет (например, иммунитет, который защищает субъекта против заражения коронавирусом и/или уменьшает тяжесть состояния, вызванного заражением коронавирусом). Защитный иммунный ответ может включать образование антител и/или клеточно-опосредованный ответ. В зависимости от контекста термин «вакцина» может также относиться к суспензии или раствору иммуногена, который вводят субъекту для выработки защитного иммунитета.
Как применен в настоящем документе, термин «субъект» включает людей и других животных. Как правило, субъект представляет собой человека. Например, субъект может представлять собой взрослого, подростка, ребенка (от 2 до 14 лет), младенца (от рождения до 2 лет) или новорожденного (до 2 месяцев). В конкретных аспектах возраст субъекта составляет до 4-х месяцев или до 6-ти месяцев. В некоторых аспектах взрослые представляют собой пожилых людей в возрасте примерно 65 лет или старше или примерно 60 лет или старше. В некоторых аспектах субъект представляет собой беременную женщину или женщину, намеревающуюся забеременеть. В других аспектах субъект не представляет собой человека; например, субъект представляет собой примата, не являющегося человеком; например, павиана, шимпанзе, гориллу или макаку. В некоторых аспектах субъект может представлять собой домашнее животное, такое как собака или кошка.
Как применен в настоящем документе, термин «фармацевтически приемлемый» означает, что он одобрен регулирующим органом федерального правительства США или правительства штата или включен в фармакопею США, Европейскую фармакопею или другую общепризнанную фармакопею для применения у млекопитающих и, более конкретно, у людей. Эти композиции могут быть применены в качестве вакцин и/или антигенных композиций для индуцирования защитного иммунного ответа у позвоночных.
Вспышка SARS-CoV-2, вызывающая COVID-19, привела к глобальной пандемии. Текущее клиническое ведение инфекции SARS-CoV-2 включает профилактику, меры контроля и поддерживающую помощь. Чтобы сдержать текущую пандемию и возможные повторения в будущем, важно лучше понять этот вирус и разработать методы быстрой диагностики, терапевтические методы лечения и профилактические вакцины для борьбы с такими опасными патогенами. Большая часть усилий по разработке вакцины и антител в основном сосредоточена на сильно гликозилированном S-белке SARS-CoV-2, важном посреднике проникновения вируса в клетку-хозяин путем связывания с рецептором ангиотензинпревращающего фермента 2 (ACE2) на поверхности клетки-хозяина. Как и многие другие вирусные слитые белки, S-белок SARS-CoV-2 использует гликановую оболочку для защиты остова S-белка как в конформации до слияния, так и после слияния и уклоняется от иммунного ответа хозяина. Однако до сих пор неизвестно, как посттрансляционная модификация влияет на транслируемый иммуноген после вакцинации мРНК, и среди событий посттрансляционной модификации гликозилирование играет важную роль в регуляции фолдинга, структуры и функции белка. Настоящее раскрытие направлено на разработку моно-GlcNAc-декорированных и сконструированных с помощью сайтов гликозилирования (удаление несущественного сайта гликозилирования с помощью обратной генетики для замены Asn на Gln) вариантов полноразмерного белка S и его субъединиц, включая S1 или S2, и домен RBD в качестве вакцин-кандидатов для исследований иммунизации с целью получения антиген-специфических нейтрализующих антител.
Считается, что разработка инновационных стратегий и защитных вакцин в широком смысле для борьбы с CoV-инфекцией может привести к важным открытиям, имеющим медицинское значение, но не подчеркивалось иное. Принципы и стратегии, разработанные в настоящем описании, обеспечивают универсальные вакцины на основе коронавирусной мРНК против различных CoV и их вариантов.
Иммуногенный пептид, полученный из шиповидного белка коронавируса
На сегодняшний день известно более 8 миллионов последовательностей белка S с более чем 1000 участками мутации в его 1273 аминокислотных последовательностях, включая высококонтагиозный мутант D614G, а также мутант из Великобритании и Южной Африки. Кроме того, все сайты гликозилирования на S-белке высококонсервативны, а консервативные пептидные эпитопы на S-белке в значительной степени экранированы гликанами. Это представляет серьезную проблему в разработке широко эффективных антител и вакцин для борьбы с новыми вирусными штаммами. В настоящем изобретении разработана более эффективная стратегия конструирования вакцины с применением S-белка со сконструированным гликозилированием в качестве иммуногенов для лучшего экспонирования высококонсервативных эпитопов для конструирования вакцины, чтобы вызывать широкие защитные иммунные ответы.
Настоящее раскрытие показало, что удаление гликановых экранов на вирусных поверхностных гликопротеинах для воздействия на более консервативные эпитопы представляет собой очень эффективный подход к разработке вакцины против SARS-CoV-2. Поскольку один остаток GlcNAc, связанный с Asn, представляет собой минимальный компонент N-гликана, необходимого для сворачивания и стабилизации гликопротеина, предполагается, что обрезка N-гликанов, чтобы оставить один GlcNAc на S-белке SARS-CoV-2, не повлияет его складывание, но будет способствовать максимальному раскрытию белкового остова, чтобы вызвать сильный и специфичный к белку иммунный ответ при сохранении его структурной целостности.
Путем удаления гликановых экранов на шиповидном белке SARS-CoV-2 настоящее изобретение обеспечивает иммуногенный пептид, включающий, по меньшей мере, одну аминокислотную последовательность, выбранную из группы, состоящей из: TESIVRFPNITNL (SEQ ID NO:41), NITNLCPFGEVFNATR (SEQ ID NO:42), LYNSASFSTFK (SEQ ID NO:43), LDSKVGGNYN (SEQ ID NO:44), KSNLKPFERDIST (SEQ ID NO:45), KPFERDISTEIYQAG (SEQ ID NO:46), GPKKSTNLVKNKC (SEQ ID NO:47), NCDVVIGIV[N]NTVY (SEQ ID NO:48), PELDSFKEELDKYFK[N]HTS (SEQ ID NO:49), VNIQKEIDRLNEVA (SEQ ID NO:50), NL[N]ESLIDLQ (SEQ ID NO:51) и LGKYEQYIKWP (SEQ ID NO:52) или аминокислотной последовательности, имеющей, по меньшей мере, примерно 99%, 98%, 97%, 96%, 95% или 90% идентичности любой из SEQ ID NO: 41-52.
В некоторых воплощениях, иммуногенный пептид включает, по меньшей мере, одну аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 41-43 и 45-51.
Аминокислотная последовательность SEQ ID NO: 41-52, по отдельности или в комбинации, может быть применена в качестве антигена(антигенов), способного(способных) стимулировать иммунный ответ против коронавирусов.
Обычные способы, например, химический синтез или рекомбинантная технология, могут быть применены для получения иммуногенного пептида, как описано в настоящем документе.
Иммуногенный пептид или вектор экспрессии, способный экспрессировать иммуногенный пептид, можно смешивать с фармацевтически приемлемым носителем с образованием иммуногенной композиции. Композицию можно вводить субъекту, нуждающемуся в этом, для профилактики или лечения коронавирусной инфекции.
Композиция может быть составлена с фармацевтически приемлемым носителем, таким как фосфатно-солевой буфер, раствор бикарбоната и/или адъювант. Подходящие фармацевтические носители и разбавители, а также необходимые фармацевтические средства для их применения известны в данной области техники. Эта композиция может быть приготовлена в виде инъекций, жидкого раствора, эмульсии или другой подходящей композиции.
Примеры адъювантов включают, но не ограничиваются ими, преципитат квасцов, полный адъювант Фрейнда, неполный адъювант Фрейнда, CpG, QS21, адъювант монофосфорил-липид A/трегалоза дикориномиколат, эмульсию вода в масле, содержащую Corynebacterium parvum и тРНК, и другие вещества, которые выполняют задачу усиления иммунного ответа, имитируя определенные наборы эволюционно консервативных молекул, включая липосомы, липополисахариды (LPS), молекулярные контейнеры для антигена, компоненты стенок бактериальных клеток и эндоцитированные нуклеиновые кислоты, такие как двухцепочечная РНК, одноцепочечная ДНК, и неметилированная ДНК, включающая динуклеотид CpG. Другие примеры включают холерный токсин, термолабильный энтеротоксин E. coli, липосомы, иммуностимулирующий комплекс (ISCOM), иммуностимулирующие последовательности олигодезоксинуклеотидов и гидроксид алюминия. Композиция также может включать полимер, облегчающий доставку in vivo.
Коронавирусные мРНК-вакцины
Коронавирусы (CoV) заражают людей и животных и вызывают различные заболевания, включая респираторные, кишечные, почечные и неврологические заболевания. CoV использует свой шиповидный гликопротеин (S), основную мишень для нейтрализующих антител, для связывания своего рецептора и обеспечения слияния мембран и проникновения вируса. Спайковый белок коронавируса высоко консервативен среди всех коронавирусов человека (CoV) и участвует в распознавании рецепторов, прикреплении вируса и проникновении в клетки-хозяева. Точно так же S-белок SARS-CoV-2 также высоко консервативен по сравнению с CoV. S-белок SARS-CoV-2 имеет три основных иммуногенных домена: N-концевой домен (NTD), домен связывания рецептора (RBD) и домен субъединицы 2 (S2). Предыдущие исследования показали, что нейтрализующие антитела (NAb), которые распознают RBD, обладают высокой степенью защиты от SARS-CoV-2 и других коронавирусов, а белок S сильно гликозилирован (24 сайта гликозилирования на мономер) и часто мутирует с миллионами последовательностей, о которых сообщает GISAID. Наиболее консервативные области S-белка SARS-CoV-2 расположены в доменах RBD и S2, которые в значительной степени экранированы гликанами (Han-Yi Huang, et al. Impact of glycosylation on a broad-spectrum vaccine against SARS-CoV-2. bioRxiv preprint. doi: www.biorxiv.org/content/10.1101/2021.05.25.445523v2.full), антитела, распознающие эти области, могут обеспечить широкую защиту от вариантов SARS-CoV-2 (Maximilian M Sauer. et al. Structural basis for broad coronavirus neutralization. Nat Struct Mol Biol. 28(6):478-486 (2021)1314; C.; Wang. et al. A conserved immunogenic and vulnerable site on the coronavirus spike protein delineated by cross-reactive monoclonal antibodies. Nat Commun. 12 (1):1715 (2021)). Гликозилирование антигенов-мишеней или патогенов регулировало индукцию антител, но до сих пор неизвестно, влияет ли гликозилирование на Т-клеточный ответ. Фактически трудно или невозможно экспрессировать белок S из плазмиды с делецией некоторых сайтов гликозилирования (Han-Yi Huang, et al. Impact of glycosylation on a broad-spectrum vaccine against SARS-CoV-2. bioRxiv preprint. doi: www.biorxiv.org/content/10.1101/2021.05.25.445523v2.full).
Настоящее раскрытие неожиданно показало, что применение технологии мРНК для удаления гликановых экранов для лучшего экспонирования консервативных областей представляет собой эффективную стратегию разработки вакцины широкого спектра действия. В настоящем изобретении мРНК шиповидного белка коронавируса (такого как S-белок SARS-CoV-2) с мутацией специфических сайтов гликозилирования применяют в качестве модели для иммунизации, чтобы исследовать, как мРНК с мутацией сайта гликозилирования влияет на экспрессию белка и иммунный ответ.
Соответственно, настоящее изобретение относится к молекуле модифицированной нуклеиновой кислоты, кодирующей модифицированный спайковый белок, включающий одну или несколько аминокислотных замен аспарагина (N) на глутамин (Q) в последовательностях N-связанного гликозилирования (N-X-S/T), где X представляет собой любой аминокислотный остаток, кроме пролина, а S/T обозначает сериновый или треониновый остаток.
Молекула модифицированной нуклеиновой кислоты может представлять собой мРНК или одноцепочечную или двухцепочечную ДНК и применяться в качестве иммуногена или вакцины против патогена. В одном воплощении возбудитель представляет собой CoV. Примеры CoV включают, помимо прочего, SARS-CoV, MERS-CoV и SARS-CoV-2. Примеры SARS-CoV-2 включают, помимо прочего, альфа-SARS-CoV2, бета-SARS-CoV2, гамма-SARS-CoV2, дельта-SARS-CoV2 и омикрон-SARS-CoV2 и их варианты.
По сравнению с шиповидным белком дикого типа штаммов Wuhan и дельта-штаммов (таких как SEQ ID NO: 2, 16, 18 и 20), модифицированный шиповидный белок, описанный в настоящем документе, включает одну или несколько аминокислотных делеций или добавлений в сиквонах N-связанного гликозилирования (N-X-S/T) для устранения сиквонов N-связанного гликана. Альтернативно, описанный в настоящем документе модифицированный спайковый белок включает одну или несколько аминокислотных замен S/T на аланин (А) в сайтах O-связанного гликозилирования для устранения сайтов O-связанного гликозилирования.
мРНК шиповидного белка коронавируса может быть применена в качестве вакцины против коронавируса, которая имеет мутацию одного или нескольких сайтов гликозилирования в рецептор-связывающем домене (RBD), домене субъединицы 1 (S1) или субъединицы 2 (S2) или их варианте.
Мутация CoV или его варианта, как описано в настоящем документе, может представлять собой делецию, добавление или замену. В некоторых воплощениях, мРНК шиповидного белка коронавируса имеет одну или несколько мутаций сайтов гликозилирования в RBD, S1 или S2 с одной или несколькими заменами N на Q или S/T на A или их комбинацией. Мутация сайтов N-гликозилирования заключается в изменении предполагаемой последовательности N-X-S/T на Q-X-S/T и/или изменении S/T на A в сайте О-гликозилирования.
Сайты гликозилирования с заменой N на Q включают, но не ограничиваются ими, следующие:
S-(deg-RBD), который представляет собой белок S, в котором все 2 N-сайты гликозилирования в RBD мутированы с N на Q, а 2 O-сайты гликозилирования мутированы с S/T на A (например, SEQ ID NO: 4, 22, 24 или 26);
S-(deg-S2), который представляет собой белок S со всеми 9 сайтами гликозилирования в S2, мутировавшими с N на Q (например, SEQ ID NO: 6, 28, 30 или 32);
S-(deg-S2-1194, который представляет собой белок S с 8 сайтами гликозилирования в S2, мутировавшими с N на Q, за исключением сайта гликозилирования 1194) (например, SEQ ID NO: 8 или 34);
S-(deg-RBD-801), который представляет собой белок S, в котором все 2 N-сайты гликозилирования в RBD мутированы с N на Q, а 2 O-сайты гликозилирования мутированы с S/T на A, а сайт гликозилирования 801 мутирован с N на Q (например, SEQ ID NO: 10 или 36);
S-(deg-RBD-1194), который представляет собой белок S, в котором все 2 N-сайты гликозилирования в RBD мутированы с N на Q, а 2 O-сайты гликозилирования мутированы с S/T на A, а сайт гликозилирования 1194 мутирован с N на Q (например, SEQ ID NO: 12 или 38); и
S-(deg-RBD-122-165-234), который представляет собой белок S, в котором все 2 N-сайты гликозилирования в RBD мутированы с N на Q, а 2 O-сайты гликозилирования мутированы с S/T на A, а сайты гликозилирования 122, 165 и 234 мутировали с N на Q (например, SEQ ID NO: 14 или 40).
В дополнительном воплощении, мРНК или ДНК для S-(deg-RBD) имеет последовательность SEQ ID NO: 3, 21, 23 или 25, мРНК или ДНК для S-(deg-S2) имеет последовательность SEQ ID NO: 5, 27 , 29 или 31, мРНК или ДНК для S-(deg-S2-1194) имеет последовательность SEQ ID NO: 7 или 33, мРНК или ДНК для S-(deg-RBD-801) имеет последовательность SEQ ID NO: 9 или 35, мРНК или ДНК для S-(deg-RBD-1194) имеет последовательность SEQ ID NO: 11 или 37, мРНК или ДНК для S-(deg-RBD-122-165-234) имеет последовательность SEQ ID NO: 13 или 39.
В настоящем изобретении предложена линейная ДНК, включающая промотор, 5'-нетранслируемую область, 3'-нетранслируемую область, экспрессионную плазмиду с S-2P или без нее и сигнальную последовательность поли(А)-хвоста, в которой предполагаемая последовательность N-X-S/T заменена на Q-X-S/T, а сайт О-гликозилирования был заменен с S/T на A на экспрессионной плазмиде. В одном воплощении, экспрессионная плазмида S-2P включает ген S вируса SARS-CoV-2, кодирующий состояние перед слиянием S, имеющего пролиновые замены K968 и V969.
мРНК может быть получена трансляцией in vitro из вышеупомянутой ДНК с применением вектора, содержащего модифицированную молекулу нуклеиновой кислоты, и клетки-хозяина, содержащей вектор, как описано в настоящем документе. Ген целевого шиповидного белка изготавливают синтетическим путем и вставляют в плазмиду или небольшой кольцевой фрагмент ДНК. Плазмиды применяют в производстве мРНК-вакцин, потому что их легко реплицировать (копировать) и они надежно содержат последовательность целевого гена. Две нити плазмидной ДНК разделяются. Затем РНК-полимераза, молекула, транскрибирующая РНК из ДНК, использует ген спайкового белка для создания одной молекулы мРНК. Наконец, другие молекулы расщепляют остальную часть плазмиды, чтобы в качестве вакцины была упакована только мРНК. Скорость и эффективность этого процесса позволяют производить большое количество мРНК за короткий период времени.
В настоящем изобретении было обнаружено, что иммунизация S-белком дикого типа с гликанами на всех N-сайтах гликозилирования, урезанными до N-ацетилглюкозамина (GlcNAc), в качестве моно-GlcNAc-декорированного S-белка (Smg) индуцировала антитело широкой защитой и CD4+, а также CD8+ Т-клеточные ответы против рассматриваемых вариантов, включая варианты альфа, бета, гамма, дельта и омикрон. Дальнейшие исследования показывают, что большинство консервативных эпитопов на белке S расположены в RBD и домене HR2 субъединицы S2, но эти консервативные эпитопы в значительной степени защищены гликанами, чтобы ускользнуть от иммунного ответа. Таким образом, удаление экранированных гликанов экспонирует более консервативные эпитопы и вызовет более широкий и сильный иммунный ответ. В настоящем изобретении также применяют технология одиночных В-клеток для скрининга В-клеток от мышей, иммунизированных Smg, для идентификации широко нейтрализующего моноклонального антитела, которое нацелено на высококонсервативную область в RBD, которая не индуцируется при иммунизации полностью гликозилированным белком S, дополнительно демонстрируя, что удаление гликановых экранов из S-белка представляет собой эффективную стратегию разработки вакцины с широким защитным действием против вариантов SARS-CoV-2. Чтобы применить этот вывод к дизайну мРНК-вакцины, в настоящем документе мы сосредоточились на изучении мРНК шиповидного белка SARS-CoV-2 с мутацией специфических сайтов гликозилирования в RBD, S1 или S2 или их комбинации с заменой N на Q и S/T на A и исследовании экспрессии их белков и иммунного ответа, а также степени защиты.
Иммунизация такой мРНК приводит к накоплению неправильно свернутого шиповидного белка в эндоплазматическом ретикулуме и вызывает активацию BiP/GRP78, XBP1 и p-eIF2α, вызывая клеточный апоптоз и CD8 Т-клеточный ответ. Кроме того, дендритные клетки (DC), инкубированные с мРНК-вакциной с делецией сайта гликозилирования в S2, увеличивали экспрессию главного комплекса гистосовместимости класса I (MHC I). Кроме того, удаление сайтов гликозилирования, влияющих на стабильность шиповидного белка, снижает выработку антител и увеличивает ответ CD8+ Т-клеток. В настоящем изобретении предложены мРНК-вакцины широкого спектра действия, которые не могут быть получены с применением экспрессированных белков в качестве антигенов.
Нанокластер мРНК и наночастицы
В одном аспекте настоящее изобретение относится к нанокластеру мРНК, содержащему мРНК-вакцину, описанную в настоящем документе, в составе липидной наночастицы.
В качестве липидной наночастицы можно применять биоразлагаемую липидную наночастицу. В одном воплощении биоразлагаемая липидная наночастица представляет собой полимеры на основе гуанидина.
В другом воплощении настоящее изобретение относится к нанокластеру мРНК, включающему биоразлагаемые липидные наночастицы, инкапсулированные с мРНК-вакциной, описанной в настоящем документе, в которой биоразлагаемые липидные наночастицы содержат гуанидиновые и цвиттерионные звенья, в которых группы на основе гуанидина, а также цвиттерионные группы присоединяются к липидному хвосту полимера, и в которых группы на основе гуанидина прикрепляются к мРНК, тем самым образуя солевые мостики между группами гуанидиния и фосфатами в мРНК. Примеры полимеров на основе гуанидина включают, но не ограничиваются ими, P1, P2, P3, Pb и Pz, как описано в настоящем документе.
В описании обеспечены липидные наночастицы на основе гуанидина в качестве носителя для композиции мРНК-нановакцины. Полимеры генерируют эффективную доставку мРНК к антигенпрезентирующим клеткам, демонстрируя сильную способность к эндосомальному высвобождению. Своевременная деградация поли(дисульфидов) внутриклеточным глутатионом также сводит к минимуму цитотоксичность по сравнению с другими неразлагаемыми наноносителями.
В другом воплощении нанокластер мРНК имеет соотношение наночастиц/мРНК (N/P), равное примерно 10 или примерно 20.
мРНК-вакцина против коронавируса по настоящему изобретению также может быть присоединена к наночастице.
Нанокластер мРНК и наночастицы представляют собой частицы размером от 1 до 100 нанометров (нм), которые можно применять в качестве субстрата для иммобилизации лигандов. Наночастица может представлять собой, например, липидную наночастицу, полимерную наночастицу, неорганическую наночастицу, такую как наночастица золота, липосома, иммуностимулирующий комплекс (ISCOM), вирусоподобная частица (VLP) или самособирающийся белок.
В одном воплощении, композиции липидных наночастиц обычно содержат один или несколько липидов. В некоторых воплощениях, липид представляет собой катионный или ионизируемый липид. В некоторых воплощениях, композиции липидных наночастиц (LNP) дополнительно содержат другие компоненты, включая фосфолипид, структурный липид, соединение четвертичного амина и молекулу, способную уменьшать агрегацию частиц, например, ПЭГ или ПЭГ-модифицированный липид.
Описанная в настоящем документе мРНК-вакцина может быть инкапсулирована в липосому или полимерсому.
Обычная липосома изготавливается путем формирования липидной бимолекулярной мембраны в одну стадию, поэтому обычно как внутреннюю, так и внешнюю мембрану липосомы изготавливают из одного и того же составляющего компонента. Примеры ингредиента липосомы включают, но не ограничиваются ими, DSPC, DOTAP, DMG, пегилированный DMG, холестерин и их комбинация. В одном воплощении мРНК-липосому получают путем смешивания мРНК и липидного ингредиента в соотношении, как описано в настоящем документе, при комнатной температуре.
Полимерсомы, как описано в настоящем документе, представляют собой оболочки, самособирающиеся из амфифильных блок-сополимеров. Эти амфифильные блок-сополимеры представляют собой макромолекулы, включающие, по меньшей мере, один блок гидрофобного полимера и, по меньшей мере, один блок гидрофильного полимера. При гидратации эти амфифильные блок-сополимеры самособираются в оболочки, так что гидрофобные блоки имеют тенденцию связываться друг с другом, чтобы свести к минимуму прямое воздействие воды и образовывать внутреннюю поверхность оболочки, а гидрофильные блоки обращены наружу, образуя внешнюю поверхность корпуса. Гидрофобное ядро этих водорастворимых полимерсом может обеспечивать среду для солюбилизации дополнительных гидрофобных молекул. Как таковые, эти водорастворимые полимерсомы могут действовать как полимеры-носители для гидрофобных молекул, инкапсулированных внутри полимерсом. Более того, самосборка амфифильных блок-полимеров происходит в отсутствие стабилизаторов, которые в противном случае обеспечивали бы коллоидную стабильность и предотвращали агрегацию. В одном воплощении липосомы мРНК получают путем смешивания мРНК и полимера в соотношении, как описано в настоящем документе, при комнатной температуре.
Вакцина, комбинированная вакцина, вакцинная композиция, способы и терапевтическое применение
Описанная в настоящем документе мРНК может быть применена в качестве вакцины либо отдельно, либо в комбинации с другими вакцинами. Соответственно, настоящее изобретение обеспечивает комбинированную вакцину, содержащую мРНК-вакцину по настоящему изобретению и одну или несколько дополнительных вакцин. Дополнительная вакцина выбрана из одной или нескольких вакцин против COVID-19, вакцины против гриппа, адвеновирусной вакцины, вакцины против сибирской язвы, вакцины против холеры, вакцины против дифтерии, вакцины против гепатита А или В, вакцины против ВПЧ, вакцины против кори, вакцины против эпидемического паротита, вакцины против оспы, ротавирусной вакцины, противотуберкулезной вакцины, пневмококковой вакцины и вакцины против Haemophilus influenzae типа b и любой их комбинации.
Настоящее раскрытие также относится к вакцинной композиции, включающей мРНК-вакцину, нанокластер мРНК или наночастицу мРНК, как описано в настоящем документе. В настоящем изобретении также обеспечен способ профилактики или лечения коронавирусной инфекции, включающий введение субъекту мРНК-вакцины, нанокластера мРНК или наночастицы мРНК или композиции вакцины, как описано в настоящем документе. В одном воплощении субъект инфицирован или подвержен риску заражения коронавирусом.
Вакцину мРНК, нанокластер мРНК или наночастицу мРНК или композицию вакцины, как описано в настоящем документе, можно вводить в виде начальной дозы и двух, трех или четырех бустерных доз. В некоторых воплощениях мРНК-вакцину, нанокластер мРНК или наночастицу мРНК или вакцинную композицию вводят в начальной дозе и, по меньшей мере, в одной бустерной дозе в течение примерно одного месяца, примерно двух месяцев, примерно трех месяцев, примерно четырех месяцев, примерно пяти месяцев или примерно через шесть месяцев после первоначальной дозы. В некоторых воплощениях обеспечиваемую композицию вводят во второй бустерной дозе приблизительно через шесть месяцев, приблизительно через семь месяцев, приблизительно через восемь месяцев, приблизительно через девять месяцев, приблизительно через десять месяцев, приблизительно через одиннадцать месяцев или приблизительно через год после введения начальной дозы.
Вакцину мРНК, нанокластер мРНК или наночастицу мРНК или вакцинную композицию вводят в виде одной или нескольких доз. В одном воплощении доза может включать или исключать от 5 мкг до 50 мкг. В некоторых воплощениях доза составляет примерно 5 мкг, 10 мкг, 15 мкг, 20 мкг, 25 мкг, 30 мкг, 35 мкг, 40 мкг, 45 мкг или 50 мкг.
Вакцинная композиция предпочтительно включает фармацевтически приемлемую вакцину, носитель или разбавитель. Вакцинная композиция может быть получена любым подходящим способом. Композиция со стандартными фармацевтически приемлемыми носителями и/или вспомогательными веществами может быть осуществлена с применением обычных методов в области фармацевтики. Точная природа препарата будет зависеть от нескольких факторов, включая вводимую вакцину и желаемый способ введения. Подходящие типы композиций полностью описаны в руководстве Remington's Pharmaceutical Sciences, 19th Edition, Mack Publishing Company, Eastern Pennsylvania, USA.
Композицию вакцины или фармацевтическую композицию, как описаны в настоящем документе, можно вводить любым путем. Подходящие пути введения включают, но не ограничиваются ими, назальный, внутривенный, внутримышечный, внутрибрюшинный, подкожный, внутрикожный, трансдермальный и пероральный/буккальный пути введения.
Композиции могут быть приготовлены вместе с физиологически приемлемым носителем или разбавителем. Обычно такие композиции готовят в виде жидких суспензий наночастиц. Наночастицы могут быть смешаны с наполнителем, который представляет собой фармацевтически приемлемый и совместимый с активным ингредиентом наполнитель. Подходящие вспомогательные вещества представляют собой, например, воду, физиологический раствор, декстрозу, глицерин и т.п. и их комбинации.
Подходящие композиции, в которых носитель представляет собой жидкость, для введения, например, в виде назального спрея или назальных капель, включают водные или масляные растворы активного ингредиента. Препараты, подходящие для аэрозольного введения, могут быть приготовлены в соответствии с обычными способами и могут быть доставлены с другими терапевтическими средствами.
Кроме того, при желании фармацевтические композиции могут содержать незначительные количества вспомогательных веществ, таких как смачивающие или эмульгирующие средства и/или рН-буферные средства.
Без намерения ограничить объем изобретения ниже приведены примеры приборов, устройств, способов и связанных с ними результатов согласно воплощениям настоящего изобретения. Обратите внимание, что в примерах могут быть применены заголовки или подзаголовки для удобства читателя, что никоим образом не должно ограничивать объем изобретения. Кроме того, в настоящем документе предлагают и раскрывают определенные теории; однако они никоим образом, правы они или нет, не должны ограничивать объем изобретения, пока изобретение осуществляется в соответствии с изобретением без учета какой-либо конкретной теории или схемы действия.
Примеры
Материалы и методы
Клеточные линии. Клетки эмбриональной почки человека (HEK293) поддерживали в модифицированной Дульбекко среде Игла (DMEM) («Invitrogen», Rockville, MD) с 10% термоинактивированной фетальной бычьей сывороткой (FBS) («Thermo Scientific») и антибиотиками (100 ед./мл пенициллина G и 100 мкг/мл стрептомицина).
Антитела и белки. Кроличье поликлональное антитело S против SARS-CoV-2 и полноразмерные белки S, S2, RBD и варианты SARS-CoV-2 (экспрессированные клетками 293T) были приобретены у «Sino Biologicals» (Пекин, Китай). Мышиные моноклональные антитела против β-актина, GAPDH и кроличьи моноклональные антитела против MHCII были приобретены у «Millipore». Кроличья моноклональная анти-Na/K-АТФаза была получена от «ABcan». Моноклональные антитела мыши против SERCA2 и моноклональные антитела кролика против MHC I были получены от «Invitrogen». Кроличьи моноклональные антитела против BiP/GRP78, XBP1 и p-eIF2α были приобретены у «ABclonal». Все коммерческие антитела были проверены на специфичность компаниями и нами с помощью вестерн-блоттинга. Для получения дегликозилированного белка белок S, RBD, S1 или S2 дегликозилировали в буферном растворе с помощью PNGase F («Sigma») при 37°С в течение 24 часов в темноте. После дегликозилирования образцы очищали и проверяли вестерн-блоттингом.
мРНК-вакцина из дегликозилированного белка S и композиция. Состояние до слияния S, кодон-оптимизированный ген S SARS-CoV-2 был синтезирован с помощью GenScript и клонирован в pcDNA3.1 или pVax, а в одном воплощении был стабилизирован заменами пролина K968 и V969 (S-2P). Была сконструирована растворимая версия S, оканчивающаяся глутамином Q1208 S-2P, за которым следовал мотив тримеризации фибрина (фолдона) T4, сайт расщепления тромбином и метка 6xHis на С-конце. Чтобы мутировать N-сайты гликозилирования, предполагаемая последовательность N-X-S/T была изменена на Q-X-S/T, а сайт О-гликозилирования был изменен с S/T на A с помощью сайт-направленного мутагенеза на экспрессионной плазмиде S-2P. Для получения мРНК-вакцины линейную ДНК, содержащую промотор Т7, 5'-нетранслируемую область, 3'-нетранслируемую область, S-2P и сигнальную последовательность поли(А)-хвоста, амплифицировали с применением ДНК-полимеразы TOOLS Ultra High Fidelity (BIOTOOLS Co., Ltd., Тайбэй, Тайвань) с 1 мкл матрицы ДНК в наборе mMESSAGE mMACHINE® Kit («Thermo Scientific») при 37°C в течение 1-го часа в соответствии с протоколом производителя. мРНК очищали с помощью набора для очистки РНК («BioLabs») в соответствии с протоколом производителя и хранили при температуре -80°С до дальнейшего применения. Для композиции мРНК-LNP, мРНК инкапсулировали в LNP с применением процесса самосборки, в котором водный раствор мРНК при рН 4,0 быстро смешивали с этанольной смесью липидов, содержащей ионизируемый катионный липид, фосфатидилхолин, холестерин и полиэтиленгликоль-липид. В композицию LNP входили DSPC («Sigma»), холестерин («Sigma»), DOTAP («Sigma») и DMG-PEG 2000 («Sigma»). мРНК-LNP охарактеризовали и впоследствии хранили при -80°С в концентрации 1 мг/мл. После трансфекции клеток HEK293 10 мкг мРНК-LNP в шести лунках планшета, через 48 часов собирали общий клеточный лизат для мониторинга экспрессии S с помощью вестерн-блоттинга.
Животные и иммунизация. Мышей BALB/c в возрасте 6-8 недель (n = 5) иммунизировали внутримышечно 50 мкг мРНК-LNP в PBS с 300 мМ сахарозы. Животных иммунизировали на 0-й неделе, ревакцинировали второй вакцинацией на 2-й неделе, а образцы сыворотки и селезенки собирали у каждой мыши через одну неделю после ревакцинации. Эксперименты на животных были оценены и одобрены Институциональным комитетом по содержанию и применению лабораторных животных Academia Sinica.
Измерение титра IgG в сыворотке. Для определения титра IgG применяли ELISA против белка S. Планшеты покрывали 50 нг/лунку варианта белка S, как показано на фиг. 4 и 5, а затем перекрыли 5% обезжиренным молоком. Последовательно добавляли сыворотку иммунизированных мышей и вторичное антитело, конъюгированное с HRP. Применяли раствор субстрата пероксидазы (TMB) и стоп-раствор 1M H2SO4, и оптическую плотность (OD 450 нм) считывали с помощью устройства для считывания микропланшетов.
Анализ нейтрализации псевдовируса для исследования сыворотки. Псевдовирус был сконструирован в профильном центре RNAi в Academia Sinica. Вкратце, псевдотипированный лентивирус, несущий S-белок SARS-CoV-2 или его вариант, получали путем временной трансфекции клеток HEK-293T с помощью pCMV-ΔR8.91, pLAS2w.Fluc. Ppuro и pcDNA3.1-nCoV-SΔ18. Клетки HEK-293T высевали за сутки до трансфекции с последующей доставкой плазмид в клетки с помощью реагента для трансфекции TransITR-LT1 («Mirus»). Культуральную среду обновляли через 16 часов и собирали через 48 и 72 часа после трансфекции. Клеточный дебрис удаляли центрифугированием, а супернатант пропускали через шприцевой фильтр 0,45 мкм («Pall Corporation»). Псевдотипированный лентивирус затем хранили при -80°C. Для оценки титра лентивируса с помощью анализа AlarmaBlue («Thermo Scientific») определяли единицу трансдукции (TU) псевдотипированного лентивируса с помощью анализа жизнеспособности клеток. Клетки HEK-293T, экспрессирующие ген ACE2 человека, высевали на 96-луночный планшет за один день до трансдукции лентивируса. Для определения титра псевдотипированного лентивируса в культуральную среду, содержащую полибрен (конечная концентрация 8 мкг/мл) («Sigma») добавляли различные количества лентивируса и проводили спиновое заражение при 1100 мкг в 96-луночном планшете в течение 30 мин. при 37°С. После инкубации в течение 16 ч культуральную среду, содержащую вирус и полибрен, удаляли и заменяли свежей полной DMEM, содержащей 2,5 мкг/мл пуромицина («Sigma»). После обработки пуромицином в течение 48 ч культуральную среду удаляли и определяли жизнеспособность клеток с помощью реагентов AlarmaBlue в соответствии с инструкцией производителя. Выживаемость неинфицированных клеток принимали за 100%, а титр вируса определяли путем построения графика выживаемости клеток в зависимости от разбавленной дозы вируса.
Для анализа нейтрализации инактивированные нагреванием сыворотки или антитела серийно разбавляли до желаемого разведения и инкубировали с 1000 ТЕ псевдотипированного лентивируса SARS-CoV-2 в среде DMEM в течение 1-го часа при 37°C. Затем в смесь в 96-луночном планшете инокулировали 10000 клеток HEK-293T, стабильно экспрессирующих человеческий ген ACE2. Через 16 часов после инфицирования культуральную среду заменяли свежей полной DMEM (с добавлением 10% FBS и 100 ед./мл пенициллина/стрептомицина) и непрерывно культивировали в течение еще 48 часов. Уровень экспрессии гена люциферазы определяли с применением системы анализа люциферазы Bright-Glo™ («Promega»). Относительную единицу освещенности (RLU) определяли с помощью Tecan i-control («Infinite 500»). Процент ингибирования рассчитывали как отношение снижения RLU в присутствии разбавленной сыворотки к значению RLU контроля без сыворотки, и формула расчета приведена ниже: (RLU контроль - RLU Сыворотка) / RLU контроль.
Информационный анализ S-белка SARS-CoV-2. 1 117 474 последовательности S-белка SARS-CoV-2 и их варианты были извлечены из базы данных Глобальной инициативы по совместному использованию данных по птичьему гриппу (версия GISAID: 18 апреля 2021 г.). Трехмерная модель структуры S-белка с репрезентативным профилем гликанов была построена с помощью программ CHARMM-GUI и OpenMM. В этом исследовании применяли трансмембранную область шиповидного белка, определенная UniProt. Ввод CHARMM-GUI включает файл PDB 6VSB_1_1_1, репрезентативный профиль гликанов и настройки параметров. Относительную доступность растворителя (RSA) шиповидного белка с репрезентативными гликанами и без них рассчитывали с помощью программы FreeSASA. Радиус зонда 7,2 Å применяли в программе FreeSASA для имитации среднего размера гипервариабельных петель в определяющей комплементарность области (CDR) антитела. Значение RSA для каждого остатка, применяемого в этом исследовании, представляло собой среднее значение RSA для трех белковых цепей. Определение доступных/скрытых остатков было таким же, как и в исследовании, проведенном Kajander, T. et al.
Анализ клеток, секретирующих GrzB и IFNγ. В общей сложности 5×105 спленоцитов иммунизированных мышей рестимулировали ex vivo смесью полноразмерных пептидов S, RBD и S2 (конечная концентрация 0,1 мкг/мл на пептид), («Sino Biologicals») в тестах GrzBELISpot («R&D Systems») в соответствии с инструкцией производителя и подсчитывали пятна. Для Т-клеточного субтипирования Т-клетки CD8+ и Т-клетки CD4+ выделяли из суспензии спленоцитов с помощью набора для магнитной сеперации клеток Dynabeads Untouched Mouse CD4 and CD8 Cells («Invitrogen») в соответствии с инструкциями производителя. Затем CD4+ или CD8+ Т-клетки (1×105) повторно стимулировали 5×104 сингенных DC, происходящих из костного мозга, нагруженных полноразмерной смесью пептидов WTS S (конечная концентрация 0,1 мкг/мл) («Sino Biologicals»). Чистоту выделенных субпопуляций Т-клеток определяли с помощью проточной цитометрии для расчета количества пятен на 1×105 CD4+ или CD8+ Т-клеток. Для проточной цитометрии клетки суспендировали в FACS-буфере [2% (об./об.) FBS в PBS] с плотностью 106 клеток/мл, и в этом исследовании применяли антитело против IFNγ (abcam). Интенсивность клеточной флуоресценции анализировали с помощью FACS Canto («BD Biosciences») и программного обеспечения FCS Express 3.0.
Измерение IFNγ и других цитокинов. IFNγ, IL-2, IL-4, IL-6, IL-12 и IL-13 измеряли с помощью набора ELISA в соответствии с протоколом производителя (IFN-γ: «Boster Biological Technology Co., Ltd»; IL-2, IL-4, IL-6, IL-12 и IL-13: «R&D Systems»).
Трансфекция ДНК-плазмидой и обработка MG132. После посева клеток HEK293 в 6-луночный планшет клетки трансфицировали 3 мкг каждой плазмиды с помощью трансфекционного реагента TransIT®-LT1 («Mirus»), а затем инкубировали с 1 мкМ MG-132 («MedChemExpress») или ДМСО при 37°С в течение 24 часов. Собирали общий лизат и экспрессию варианта S анализировали вестерн-блоттингом.
Трансляция in vitro. Трансляцию in vitro выполняли с помощью плазмиды, кодирующей S-2P, с применением экспрессии гликопротеина в системе IVT человека («Thermo») в соответствии с инструкциями производителя. Экспрессию белка S в разные периоды времени инкубации контролировали с помощью набора ELISA для шиповидных белков SARS-COV-2 («ABclonal») в соответствии с протоколом производителя.
Обнаружение ответа несвернутого белка. После трансфекции клеток HEK293 10 мкг мРНК S с помощью набора для трансфекции TransIT®-mRNA Transfection Kit («Mirus») в течение 48 часов, плазматическую мембрану и ER выделяли с помощью набора для обогащения ER Minute™ («Invent Biotech») в соответствии с протоколом производителя. Белок S в плазматической мембране, цитозоле и ER анализировали вестерн-блоттингом. Собирали общий лизат и определяли маркеры UPR XBP1, BiP/GRP78 и p-eIF2α с помощью вестерн-блоттинга. Клетки апоптоза измеряли с помощью набора для анализа APO™-BrdU TUNEL («Thermo») в соответствии с инструкциями производителя.
Растворимая версия экспрессии S-2P. Клетки HEK293 трансфицировали 10 мкг мРНК, кодирующей растворимую версию варианта S-2P, с помощью набора TransIT®-mRNA Transfection Kit («Mirus») в течение 72 часов, белок S очищали из супернатантов клеток с применением аффинной колонки Ni-NTA («GE Healthcare»). Очищенный белок и общий лизат контролировали на уровень белка S с помощью вестерн-блоттинга.
мРНК-вакцина индуцировала экспрессию MHC I/II на DC. DC выделяли у мышей с помощью набора M-pluriBead Cell Separation (pluriSelect) в соответствии с процедурой компании и инкубировали с 10 мкг мРНК-LNP в культуральной среде ДК (RPMI 1640 с добавлением 20 нг/мл мышиного GM-CSF («R&D Systems»), 10% FBS, 50 мкМ 2-ME, 100 единиц/мл пенициллина и 100 мкг/мл стрептомицина) при 37°С в течение 48 ч, затем анализировали экспрессию MHC I и MHC II с помощью проточной цитометрии.
Статистика и воспроизводимость. Все данные были представлены как среднее ± стандартная ошибка среднего. Номера проб и повторов экспериментов указаны так, как указано в подписях к рисункам. Сравнения между группами определяли с применением t-критерия Стьюдента. Различия считались достоверными при *P < 0,001, **P < 0,05. Все данные были проанализированы с применением программного обеспечения GraphPad Prism 6.
Пример 1. Гликозилирование белка S влияет на выработку антител
В рамках наших усилий по выявлению возможных консервативных эпитопов в качестве мишеней для разработки антител и разработки вакцин следующего поколения, а также для разработки универсальной вакцины с широкими защитными иммунными реакциями, мы провели анализ мутаций белка S из 218 516 доступных последовательностей S-белка SARS-CoV-2. Белок S имеет 1273 аминокислоты, и среди 218 516 проанализированных последовательностей имеется 1149 вариабельных аминокислотных положений, в том числе 613 в домене S1 (672 аминокислоты), 524 в домене S2 (588 аминокислот) и 134 в RBD (152 аминокислоты); однако частота мутаций составляет менее 0,1% в 1076 сайтах и более 0,1% в 73 сайтах. Консервативные последовательности можно найти в областях S1, S2 и RBD, а самая длинная из R983-I1013 находится рядом с доменом HR1 в области S2. Все 22 сайта N-гликозилирования высококонсервативны среди вариантов SARS-CoV-2. Дальнейший анализ большего количества последовательностей (около 6 миллионов) показывает аналогичное распределение консервативных эпитопов, 7 из которых находятся в RBD и 5 в HR2, а 10 консервативных эпитопов экранированы гликанами (фиг. 1). Мы считаем, что удаление гликановых экранов на поверхностных гликопротеинах вируса для воздействия на более консервативные эпитопы представляет собой очень эффективный и общий подход к разработке вакцины против SARS-CoV-2. Поскольку один остаток GlcNAc, связанный с Asn, представляет собой минимальный компонент N-гликана, необходимого для сворачивания и стабилизации гликопротеина, предполагается, что такая обрезка N-гликанов, оставляющая один GlcNAc на S-белке SARS-CoV-2, не повлияет на его сворачивание, но будет способствовать максимальному раскрытию белкового остова, чтобы вызвать сильный и специфичный к белку иммунный ответ при сохранении его структурной целостности.
Основываясь на наших предварительных результатах, полностью гликозилированные (немодифицированные) и моно-GlcNAc-декорированные (по всем N-сайтам гликозилирования) полноразмерные S, S1, S2 и RBD применяют в качестве иммуногенов для исследований иммунизации. Мы производим субъединицы S, S1, S2 и RBD с сохранением основных сайтов гликозилирования, обнаруженных в нашем предварительном исследовании, как описано выше, и делецией специфических сайтов гликозилирования и их декорированных моно-GlcNAc вариантов в качестве иммуногенов для иммунизации. Антисыворотки тестируют на их взаимодействие с репрезентативными вариантами S-белка и нейтрализующую активность против инфекции, опосредованной псевдовирусом, а сыворотки с широкой защитной активностью дополнительно исследуют, включая картирование эпитопов и адъювантное действие на ответы CD4+ и CD8+ Т-клеток. Моно-GlcNAc-декорированные варианты получают путем удаления гетерогенного гликанового слоя на N-сайтах гликозилирования полноразмерных S, S1, S2 и RBD, которые производят с применением более универсальных и хорошо зарекомендовавших себя систем экспрессии клеточных линий CHO, HEK293 или Gnt1-дефицитных HEK293. Гликаны экспрессии S-белка в этих клеточных линиях могут быть обработаны эндогликозидазами для получения желаемого белка с моно-GlcNAc во всех сайтах N-гликозилирования, а белок из последнего (Gnt1-дефицитный HEK293) относится к типам с высоким содержанием маннозы и может быть расщеплен с применением эндогликозидазы H (Endo-H) для получения желаемого белка с моно-GlcNAc во всех сайтах N-гликозилирования. Поскольку О-гликаны важны для проникновения вируса, никаких модификаций не проводили; но при необходимости их можно обработать коктейлями экзогликозидаз. Этот моно-GlcNAc, декорированный полноразмерный и укороченный S-белки, изучают на предмет их структурной целостности. Иммуногены, содержащие полностью гликозилированные и моно-GlcNAc белки, а также сконструированный с помощью сайтов гликозилирования S-белок (путем замены Asn на Gln, как показано в исследовании обратной генетики), применяют для иммунизации мышей для идентификации антител, которые нацелены на различные домены S-белка с широкой нейтрализующей активностью. Специфичность сывороточных антител проверяют с помощью полностью и моно-GlcNAc декорированного, а также S-белка, сконструированного с помощью сайта гликозилирования, и его укороченных форм. Кроме того, для изучения специфичности связывания и CD8+ Т-клеточного ответа у трансгенных мышей с гуманизированным рецептором ACE2 применяют набор синтетических пептидов с декорированным моно-GlcNAc или без него или гликопептидов, полученных в результате расщепления протеазами декорированного моно-GlcNAc S-белка. Сыворотки иммунизированных мышей дополнительно оценивали на нейтрализующую активность с помощью анализов нейтрализации псевдовирусов, разработанных в нашей лаборатории. На фиг. 2 представлена идентификация N- и O-сайтов гликозилирования и мутаций в вариантах. Все 24 сайта гликозилирования высококонсервативны среди 6 миллионов последовательностей S-белка.
Белок S часто мутирует и сильно гликозилируется с помощью 22 N- и 2 O-сайтов гликозилирования (2 N- и 2 O-сайта гликозилирования в RBD и 6 сайтов N-гликозилирования в S2), чтобы избежать иммунного ответа хозяина (фиг. 16). Чтобы изучить, влияет ли и как мРНК-вакцина S-белка с мутантными сайтами гликозилирования на экспрессию S-белка и иммунный ответ, мы мутировали несколько сайтов N-гликозилирования (с N на Q в последовательности N-X-S/T) и сайтов O-гликозилирования (с S/T на А) в области RBD или S2 мРНК, соответственно. После подтверждения экспрессии варианта S-белка до сияния в клеточной линии HEK293 (фиг. 3), мРНК, которая кодирует S-белок или S-белок с мутацией сайтов гликозилирования, инкапсулировали в LNP с образованием мРНК-LNP для иммунизации мышей. Сыворотки мышей, иммунизированных мРНК со всеми мутированными N-сайтами гликозилирования S2 (S-(deg-S2)) или со всеми мутированными N-сайтами гликозилирования S2, кроме N-1194 (S-(S2-1194)), показали меньший титр IgG против полностью гликозилированный белок S WT (фиг. 4A), S2 (фиг. 4B), RBD (фиг. 4C) или дегликозилированный белок S (фиг. 4D), но имел более высокий титр IgG против дегликозилированного антигена S2 (фиг. 4E) в анализе ELISA по сравнению с немодифицированной мРНК. Однако мыши, иммунизированные мРНК со всеми делетированными сайтами гликозилирования RBD (S-(deg-RBD)) вызывали несколько меньший титр IgG против полностью гликозилированного RBD, но более высокий титр IgG для распознавания дегликозилированного антигена RBD (фиг. 4F), предполагая, что гликозилирование S-белка влияет на продукцию антител и специфичность их связывания. Кроме того, иммунизация S-(deg-RBD), S-(deg-S2) или S-(S2-1194) мРНК вызывала более высокий титр IgG против альфа (фиг. 5A), бета (фиг. 5B), гамма (фиг. 5C) дельта и варианты омикрон (фиг. 5D), что свидетельствует о том, что гликозилирование белка S регулирует специфичность антител, генерируемых мРНК-вакциной. Чтобы проанализировать влияние мутации сайта гликозилирования на нейтрализующую активность антител, полученных от иммунизированных мышей, был проведен анализ нейтрализации псевдовируса, и результат показал, что мРНК-вакцина с делецией сайтов гликозилирования в RBD или S2 генерировала антитела со сниженной нейтрализующей активностью против псевдовируса WT (фиг. 5), но с лучшей нейтрализующей активностью в отношении четырех рассматриваемых вариантов, чем у WT (фиг. 6 и табл. 1). Чтобы лучше понять это наблюдение, анализ последовательности показал, что мутация сайтов гликозилирования в RBD или S2 обнажает более консервативные эпитопы, вызывающие иммунные ответы (фиг. 6), и что домены RBD и S2 содержат большинство высококонсервативных последовательностей в белке S (7 в RBD, 5 в HR2 S2). Эти результаты позволяют предположить, что мутация определенных сайтов гликозилирования в мРНК вакцины с S-белком будет влиять на выработку антител и их специфичность, а также на иммунный ответ.
Последовательность ДНК или РНК WT S (штамм Wuhan) (от 5'-конца к 3'-концу):
atgttcgtcttcctggtcctgctgcctctggtctcctcacagtgcgtcaatctgacaactcggactcagctgccacctgcttatactaatagcttcaccagaggcgtgtactatcctgacaaggtgtttagaagctccgtgctgcactctacacaggatctgtttctgccattctttagcaacgtgacctggttccacgccatccacgtgagcggcaccaatggcacaaagcggttcgacaatcccgtgctgccttttaacgatggcgtgtacttcgcctctaccgagaagagcaacatcatcagaggctggatctttggcaccacactggactccaagacacagtctctgctgatcgtgaacaatgccaccaacgtggtcatcaaggtgtgcgagttccagttttgtaatgatcccttcctgggcgtgtactatcacaagaacaataagagctggatggagtccgagtttagagtgtattctagcgccaacaactgcacatttgagtacgtgagccagcctttcctgatggacctggagggcaagcagggcaatttcaagaacctgagggagttcgtgtttaagaatatcgacggctacttcaaaatctactctaagcacacccccatcaacctggtgcgcgacctgcctcagggcttcagcgccctggagcccctggtggatctgcctatcggcatcaacatcacccggtttcagacactgctggccctgcacagaagctacctgacacccggcgactcctctagcggatggaccgccggcgctgccgcctactatgtgggctacctccagccccggaccttcctgctgaagtacaacgagaatggcaccatcacagacgcagtggattgcgccctggaccccctgagcgagacaaagtgtacactgaagtcctttaccgtggagaagggcatctatcagacatccaatttcagggtgcagccaaccgagtctatcgtgcgctttcctaatatcacaaacctgtgcccatttggcgaggtgttcaacgcaacccgcttcgccagcgtgtacgcctggaataggaagcggatcagcaactgcgtggccgactatagcgtgctgtacaactccgcctctttcagcacctttaagtgctatggcgtgtcccccacaaagctgaatgacctgtgctttaccaacgtctacgccgattctttcgtgatcaggggcgacgaggtgcgccagatcgcccccggccagacaggcaagatcgcagactacaattataagctgccagacgatttcaccggctgcgtgatcgcctggaacagcaacaatctggattccaaagtgggcggcaactacaattatctgtaccggctgtttagaaagagcaatctgaagcccttcgagagggacatctctacagaaatctaccaggccggcagcaccccttgcaatggcgtggagggctttaactgttatttcccactccagtcctacggcttccagcccacaaacggcgtgggctatcagccttaccgcgtggtggtgctgagctttgagctgctgcacgccccagcaacagtgtgcggccccaagaagtccaccaatctggtgaagaacaagtgcgtgaacttcaacttcaacggcctgaccggcacaggcgtgctgaccgagtccaacaagaagttcctgccatttcagcagttcggcagggacatcgcagataccacagacgccgtgcgcgacccacagaccctggagatcctggacatcacaccctgctctttcggcggcgtgagcgtgatcacacccggcaccaatacaagcaaccaggtggccgtgctgtatcaggacgtgaattgtaccgaggtgcccgtggctatccacgccgatcagctgaccccaacatggcgggtgtacagcaccggctccaacgtcttccagacaagagccggatgcctgatcggagcagagcacgtgaacaattcctatgagtgcgacatcccaatcggcgccggcatctgtgcctcttaccagacccagacaaactctcccagaagagcccggagcgtggcctcccagtctatcatcgcctataccatgtccctgggcgccgagaacagcgtggcctactctaacaatagcatcgccatcccaaccaacttcacaatctctgtgaccacagagatcctgcccgtgtccatgaccaagacatctgtggactgcacaatgtatatctgtggcgattctaccgagtgcagcaacctgctgctccagtacggcagcttttgtacccagctgaatagagccctgacaggcatcgccgtggagcaggataagaacacacaggaggtgttcgcccaggtgaagcaaatctacaagaccccccctatcaaggactttggcggcttcaatttttcccagatcctgcctgatccatccaagccttctaagcggagctttatcgaggacctgctgttcaacaaggtgaccctggccgatgccggcttcatcaagcagtatggcgattgcctgggcgacatcgcagccagggacctgatctgcgcccagaagtttaatggcctgaccgtgctgccacccctgctgacagatgagatgatcgcacagtacacaagcgccctgctggccggcaccatcacatccggatggaccttcggcgcaggagccgccctccagatcccctttgccatgcagatggcctataggttcaacggcatcggcgtgacccagaatgtgctgtacgagaaccagaagctgatcgccaatcagtttaactccgccatcggcaagatccaggacagcctgtcctctacagccagcgccctgggcaagctccaggatgtggtgaatcagaacgcccaggccctgaataccctggtgaagcagctgagcagcaacttcggcgccatctctagcgtgctgaatgacatcctgagccggctggacaaggtggaggcagaggtgcagatcgaccggctgatcaccggccggctccagagcctccagacctatgtgacacagcagctgatcagggccgccgagatcagggccagcgccaatctggcagcaaccaagatgtccgagtgcgtgctgggccagtctaagagagtggacttttgtggcaagggctatcacctgatgtccttccctcagtctgccccacacggcgtggtgtttctgcacgtgacctacgtgcccgcccaggagaagaacttcaccacagcccctgccatctgccacgatggcaaggcccactttccaagggagggcgtgttcgtgtccaacggcacccactggtttgtgacacagcgcaatttctacgagccccagatcatcaccacagacaacaccttcgtgagcggcaactgtgacgtggtcatcggcatcgtgaacaataccgtgtatgatccactccagcccgagctggacagctttaaggaggagctggataagtatttcaagaatcacacctcccctgacgtggatctgggcgacatcagcggcatcaatgcctccgtggtgaacatccagaaggagatcgaccgcctgaacgaggtggctaagaatctgaacgagagcctgatcgacctccaggagctgggcaagtatgagcagtacatcaagtggccctggtacatctggctgggcttcatcgccggcctgatcgccatcgtgatggtgaccatcatgctgtgctgtatgacatcctgctgttcttgcctgaagggctgctgtagctgtggctcctgctgtaagtttgacgaggatgactctgaacctgtgctgaagggcgtgaagctgcattacacctaa (SEQ ID NO: 1).
Белковая последовательность WT S (штамм Wuhan) (от N-конца к C-концу):
MFVFLVLLPLVSSQCVNLTTRTQLPPAYTNSFTRGVYYPDKVFRSSVLHSTQDLFLPFFSNVTWFHAIHVSGTNGTKRFDNPVLPFNDGVYFASTEKSNIIRGWIFGTTLDSKTQSLLIVNNATNVVIKVCEFQFCNDPFLGVYYHKNNKSWMESEFRVYSSANNCTFEYVSQPFLMDLEGKQGNFKNLREFVFKNIDGYFKIYSKHTPINLVRDLPQGFSALEPLVDLPIGINITRFQTLLALHRSYLTPGDSSSGWTAGAAAYYVGYLQPRTFLLKYNENGTITDAVDCALDPLSETKCTLKSFTVEKGIYQTSNFRVQPTESIVRFPNITNLCPFGEVFNATRFASVYAWNRKRISNCVADYSVLYNSASFSTFKCYGVSPTKLNDLCFTNVYADSFVIRGDEVRQIAPGQTGKIADYNYKLPDDFTGCVIAWNSNNLDSKVGGNYNYLYRLFRKSNLKPFERDISTEIYQAGSTPCNGVEGFNCYFPLQSYGFQPTNGVGYQPYRVVVLSFELLHAPATVCGPKKSTNLVKNKCVNFNFNGLTGTGVLTESNKKFLPFQQFGRDIADTTDAVRDPQTLEILDITPCSFGGVSVITPGTNTSNQVAVLYQDVNCTEVPVAIHADQLTPTWRVYSTGSNVFQTRAGCLIGAEHVNNSYECDIPIGAGICASYQTQTNSPRRARSVASQSIIAYTMSLGAENSVAYSNNSIAIPTNFTISVTTEILPVSMTKTSVDCTMYICGDSTECSNLLLQYGSFCTQLNRALTGIAVEQDKNTQEVFAQVKQIYKTPPIKDFGGFNFSQILPDPSKPSKRSFIEDLLFNKVTLADAGFIKQYGDCLGDIAARDLICAQKFNGLTVLPPLLTDEMIAQYTSALLAGTITSGWTFGAGAALQIPFAMQMAYRFNGIGVTQNVLYENQKLIANQFNSAIGKIQDSLSSTASALGKLQDVVNQNAQALNTLVKQLSSNFGAISSVLNDILSRLDPPEAEVQIDRLITGRLQSLQTYVTQQLIRAAEIRASANLAATKMSECVLGQSKRVDFCGKGYHLMSFPQSAPHGVVFLHVTYVPAQEKNFTTAPAICHDGKAHFPREGVFVSNGTHWFVTQRNFYEPQIITTDNTFVSGNCDVVIGIVNNTVYDPLQPELDSFKEELDKYFKNHTSPDVDLGDISGINASVVNIQKEIDRLNEVAKNLNESLIDLQELGKYEQYIKWPWYIWLGFIAGLIAIVMVTIMLCCMTSCCSCLKGCCSCGSCCKFDEDDSEPVLKGVKLHYT (SEQ ID NO: 2).
Последовательность ДНК или РНК WT S (штамм дельта) (от 5'-конца к 3'-концу):
atgttcgtcttcctggtcctgctgcctctggtctcctcacagtgcgtcaatctgcgaactcggactcagctgccacctgcttatactaatagcttcaccagaggcgtgtactatcctgacaaggtgtttagaagctccgtgctgcactctacacaggatctgtttctgccattctttagcaacgtgacctggttccacgccatccacgtgagcggcaccaatggcacaaagcggttcgacaatcccgtgctgccttttaacgatggcgtgtacttcgcctctatcgagaagagcaacatcatcagaggctggatctttggcaccacactggactccaagacacagtctctgctgatcgtgaacaatgccaccaacgtggtcatcaaggtgtgcgagttccagttttgtaatgatcccttcctggacgtgtactatcacaagaacaataagagctggatggagtccggagtgtattctagcgccaacaactgcacatttgagtacgtgagccagcctttcctgatggacctggagggcaagcagggcaatttcaagaacctgagggagttcgtgtttaagaatatcgacggctacttcaaaatctactctaagcacacccccatcaacctggtgcgcgacctgcctcagggcttcagcgccctggagcccctggtggatctgcctatcggcatcaacatcacccggtttcagacactgctggccctgcacagaagctacctgacacccggcgactcctctagcggatggaccgccggcgctgccgcctactatgtgggctacctccagccccggaccttcctgctgaagtacaacgagaatggcaccatcacagacgcagtggattgcgccctggaccccctgagcgagacaaagtgtacactgaagtcctttaccgtggagaagggcatctatcagacatccaatttcagggtgcagccaaccgagtctatcgtgcgctttcctaatatcacaaacctgtgcccatttggcgaggtgttcaacgcaacccgcttcgccagcgtgtacgcctggaataggaagcggatcagcaactgcgtggccgactatagcgtgctgtacaactccgcctctttcagcacctttaagtgctatggcgtgtcccccacaaagctgaatgacctgtgctttaccaacgtctacgccgattctttcgtgatcaggggcgacgaggtgcgccagatcgcccccggccagacaggcaagatcgcagactacaattataagctgccagacgatttcaccggctgcgtgatcgcctggaacagcaacaatctggattccaaagtgggcggcaactacaattatcggtaccggctgtttagaaagagcaatctgaagcccttcgagagggacatctctacagaaatctaccaggccggcagcaagccttgcaatggcgtggagggctttaactgttatttcccactccagtcctacggcttccagcccacaaacggcgtgggctatcagccttaccgcgtggtggtgctgagctttgagctgctgcacgccccagcaacagtgtgcggccccaagaagtccaccaatctggtgaagaacaagtgcgtgaacttcaacttcaacggcctgaccggcacaggcgtgctgaccgagtccaacaagaagttcctgccatttcagcagttcggcagggacatcgcagataccacagacgccgtgcgcgacccacagaccctggagatcctggacatcacaccctgctctttcggcggcgtgagcgtgatcacacccggcaccaatacaagcaaccaggtggccgtgctgtatcagggcgtgaattgtaccgaggtgcccgtggctatccacgccgatcagctgaccccaacatggcgggtgtacagcaccggctccaacgtcttccagacaagagccggatgcctgatcggagcagagcacgtgaacaattcctatgagtgcgacatcccaatcggcgccggcatctgtgcctcttaccagacccagacaaactctcgcagaagagcccggagcgtggcctcccagtctatcatcgcctataccatgtccctgggcgccgagaacagcgtggcctactctaacaatagcatcgccatcccaaccaacttcacaatctctgtgaccacagagatcctgcccgtgtccatgaccaagacatctgtggactgcacaatgtatatctgtggcgattctaccgagtgcagcaacctgctgctccagtacggcagcttttgtacccagctgaatagagccctgacaggcatcgccgtggagcaggataagaacacacaggaggtgttcgcccaggtgaagcaaatctacaagaccccccctatcaaggactttggcggcttcaatttttcccagatcctgcctgatccatccaagccttctaagcggagctttatcgaggacctgctgttcaacaaggtgaccctggccgatgccggcttcatcaagcagtatggcgattgcctgggcgacatcgcagccagggacctgatctgcgcccagaagtttaatggcctgaccgtgctgccacccctgctgacagatgagatgatcgcacagtacacaagcgccctgctggccggcaccatcacatccggatggaccttcggcgcaggagccgccctccagatcccctttgccatgcagatggcctataggttcaacggcatcggcgtgacccagaatgtgctgtacgagaaccagaagctgatcgccaatcagtttaactccgccatcggcaagatccaggacagcctgtcctctacagccagcgccctgggcaagctccagaatgtggtgaatcagaacgcccaggccctgaataccctggtgaagcagctgagcagcaacttcggcgccatctctagcgtgctgaatgacatcctgagccggctggacaaggtggaggcagaggtgcagatcgaccggctgatcaccggccggctccagagcctccagacctatgtgacacagcagctgatcagggccgccgagatcagggccagcgccaatctggcagcaaccaagatgtccgagtgcgtgctgggccagtctaagagagtggacttttgtggcaagggctatcacctgatgtccttccctcagtctgccccacacggcgtggtgtttctgcacgtgacctacgtgcccgcccaggagaagaacttcaccacagcccctgccatctgccacgatggcaaggcccactttccaagggagggcgtgttcgtgtccaacggcacccactggtttgtgacacagcgcaatttctacgagccccagatcatcaccacagacaacaccttcgtgagcggcaactgtgacgtggtcatcggcatcgtgaacaataccgtgtatgatccactccagcccgagctggacagctttaaggaggagctggataagtatttcaagaatcacacctcccctgacgtggatctgggcgacatcagcggcatcaatgcctccgtggtgaacatccagaaggagatcgaccgcctgaacgaggtggctaagaatctgaacgagagcctgatcgacctccaggagctgggcaagtatgagcagtacatcaagtggccctggtacatctggctgggcttcatcgccggcctgatcgccatcgtgatggtgaccatcatgctgtgctgtatgacatcctgctgttcttgcctgaagggctgctgtagctgtggctcctgctgtaagtttgacgaggatgactctgaacctgtgctgaagggcgtgaagctgcattacacctaa (SEQ ID NO: 15).
Белковая последовательность WT S (штамм дельта) (от N-конца к C-концу):
MFVFLVLLPLVSSQCVNLRTRTQLPPAYTNSFTRGVYYPDKVFRSSVLHSTQDLFLPFFSNVTWFHAIHVSGTNGTKRFDNPVLPFNDGVYFASIEKSNIIRGWIFGTTLDSKTQSLLIVNNATNVVIKVCEFQFCNDPFLDVYYHKNNKSWMESGVYSSANNCTFEYVSQPFLMDLEGKQGNFKNLREFVFKNIDGYFKIYSKHTPINLVRDLPQGFSALEPLVDLPIGINITRFQTLLALHRSYLTPGDSSSGWTAGAAAYYVGYLQPRTFLLKYNENGTITDAVDCALDPLSETKCTLKSFTVEKGIYQTSNFRVQPTESIVRFPNITNLCPFGEVFNATRFASVYAWNRKRISNCVADYSVLYNSASFSTFKCYGVSPTKLNDLCFTNVYADSFVIRGDEVRQIAPGQTGKIADYNYKLPDDFTGCVIAWNSNNLDSKVGGNYNYRYRLFRKSNLKPFERDISTEIYQAGSKPCNGVEGFNCYFPLQSYGFQPTNGVGYQPYRVVVLSFELLHAPATVCGPKKSTNLVKNKCVNFNFNGLTGTGVLTESNKKFLPFQQFGRDIADTTDAVRDPQTLEILDITPCSFGGVSVITPGTNTSNQVAVLYQGVNCTEVPVAIHADQLTPTWRVYSTGSNVFQTRAGCLIGAEHVNNSYECDIPIGAGICASYQTQTNSRRRARSVASQSIIAYTMSLGAENSVAYSNNSIAIPTNFTISVTTEILPVSMTKTSVDCTMYICGDSTECSNLLLQYGSFCTQLNRALTGIAVEQDKNTQEVFAQVKQIYKTPPIKDFGGFNFSQILPDPSKPSKRSFIEDLLFNKVTLADAGFIKQYGDCLGDIAARDLICAQKFNGLTVLPPLLTDEMIAQYTSALLAGTITSGWTFGAGAALQIPFAMQMAYRFNGIGVTQNVLYENQKLIANQFNSAIGKIQDSLSSTASALGKLQNVVNQNAQALNTLVKQLSSNFGAISSVLNDILSRLDKVEAEVQIDRLITGRLQSLQTYVTQQLIRAAEIRASANLAATKMSECVLGQSKRVDFCGKGYHLMSFPQSAPHGVVFLHVTYVPAQEKNFTTAPAICHDGKAHFPREGVFVSNGTHWFVTQRNFYEPQIITTDNTFVSGNCDVVIGIVNNTVYDPLQPELDSFKEELDKYFKNHTSPDVDLGDISGINASVVNIQKEIDRLNEVAKNLNESLIDLQELGKYEQYIKWPWYIWLGFIAGLIAIVMVTIMLCCMTSCCSCLKGCCSCGSCCKFDEDDSEPVLKGVKLHYT (SEQ ID NO: 16).
Последовательность ДНК или РНК WT S (штамм Wuhan S-2P) (от 5'-конца к 3'-концу):
atgttcgtcttcctggtcctgctgcctctggtctcctcacagtgcgtcaatctgacaactcggactcagctgccacctgcttatactaatagcttcaccagaggcgtgtactatcctgacaaggtgtttagaagctccgtgctgcactctacacaggatctgtttctgccattctttagcaacgtgacctggttccacgccatccacgtgagcggcaccaatggcacaaagcggttcgacaatcccgtgctgccttttaacgatggcgtgtacttcgcctctaccgagaagagcaacatcatcagaggctggatctttggcaccacactggactccaagacacagtctctgctgatcgtgaacaatgccaccaacgtggtcatcaaggtgtgcgagttccagttttgtaatgatcccttcctgggcgtgtactatcacaagaacaataagagctggatggagtccgagtttagagtgtattctagcgccaacaactgcacatttgagtacgtgagccagcctttcctgatggacctggagggcaagcagggcaatttcaagaacctgagggagttcgtgtttaagaatatcgacggctacttcaaaatctactctaagcacacccccatcaacctggtgcgcgacctgcctcagggcttcagcgccctggagcccctggtggatctgcctatcggcatcaacatcacccggtttcagacactgctggccctgcacagaagctacctgacacccggcgactcctctagcggatggaccgccggcgctgccgcctactatgtgggctacctccagccccggaccttcctgctgaagtacaacgagaatggcaccatcacagacgcagtggattgcgccctggaccccctgagcgagacaaagtgtacactgaagtcctttaccgtggagaagggcatctatcagacatccaatttcagggtgcagccaaccgagtctatcgtgcgctttcctaatatcacaaacctgtgcccatttggcgaggtgttcaacgcaacccgcttcgccagcgtgtacgcctggaataggaagcggatcagcaactgcgtggccgactatagcgtgctgtacaactccgcctctttcagcacctttaagtgctatggcgtgtcccccacaaagctgaatgacctgtgctttaccaacgtctacgccgattctttcgtgatcaggggcgacgaggtgcgccagatcgcccccggccagacaggcaagatcgcagactacaattataagctgccagacgatttcaccggctgcgtgatcgcctggaacagcaacaatctggattccaaagtgggcggcaactacaattatctgtaccggctgtttagaaagagcaatctgaagcccttcgagagggacatctctacagaaatctaccaggccggcagcaccccttgcaatggcgtggagggctttaactgttatttcccactccagtcctacggcttccagcccacaaacggcgtgggctatcagccttaccgcgtggtggtgctgagctttgagctgctgcacgccccagcaacagtgtgcggccccaagaagtccaccaatctggtgaagaacaagtgcgtgaacttcaacttcaacggcctgaccggcacaggcgtgctgaccgagtccaacaagaagttcctgccatttcagcagttcggcagggacatcgcagataccacagacgccgtgcgcgacccacagaccctggagatcctggacatcacaccctgctctttcggcggcgtgagcgtgatcacacccggcaccaatacaagcaaccaggtggccgtgctgtatcaggacgtgaattgtaccgaggtgcccgtggctatccacgccgatcagctgaccccaacatggcgggtgtacagcaccggctccaacgtcttccagacaagagccggatgcctgatcggagcagagcacgtgaacaattcctatgagtgcgacatcccaatcggcgccggcatctgtgcctcttaccagacccagacaaactctcccagaagagcccggagcgtggcctcccagtctatcatcgcctataccatgtccctgggcgccgagaacagcgtggcctactctaacaatagcatcgccatcccaaccaacttcacaatctctgtgaccacagagatcctgcccgtgtccatgaccaagacatctgtggactgcacaatgtatatctgtggcgattctaccgagtgcagcaacctgctgctccagtacggcagcttttgtacccagctgaatagagccctgacaggcatcgccgtggagcaggataagaacacacaggaggtgttcgcccaggtgaagcaaatctacaagaccccccctatcaaggactttggcggcttcaatttttcccagatcctgcctgatccatccaagccttctaagcggagctttatcgaggacctgctgttcaacaaggtgaccctggccgatgccggcttcatcaagcagtatggcgattgcctgggcgacatcgcagccagggacctgatctgcgcccagaagtttaatggcctgaccgtgctgccacccctgctgacagatgagatgatcgcacagtacacaagcgccctgctggccggcaccatcacatccggatggaccttcggcgcaggagccgccctccagatcccctttgccatgcagatggcctataggttcaacggcatcggcgtgacccagaatgtgctgtacgagaaccagaagctgatcgccaatcagtttaactccgccatcggcaagatccaggacagcctgtcctctacagccagcgccctgggcaagctccaggatgtggtgaatcagaacgcccaggccctgaataccctggtgaagcagctgagcagcaacttcggcgccatctctagcgt
gacctatgtgacacagcagctgatcagggccgccgagatcagggccagcgccaatctggcagcaaccaagatgtccgagtgcgtgctgggccagtctaagagagtggacttttgtggcaagggctatcacctgatgtccttccctcagtctgccccacacggcgtggtgtttctgcacgtgacctacgtgcccgcccaggagaagaacttcaccacagcccctgccatctgccacgatggcaaggcccactttccaagggagggcgtgttcgtgtccaacggcacccactggtttgtgacacagcgcaatttctacgagccccagatcatcaccacagacaacaccttcgtgagcggcaactgtgacgtggtcatcggcatcgtgaacaataccgtgtatgatccactccagcccgagctggacagctttaaggaggagctggataagtatttcaagaatcacacctcccctgacgtggatctgggcgacatcagcggcatcaatgcctccgtggtgaacatccagaaggagatcgaccgcctgaacgaggtggctaagaatctgaacgagagcctgatcgacctccaggagctgggcaagtatgagcagtacatcaagtggccctggtacatctggctgggcttcatcgccggcctgatcgccatcgtgatggtgaccatcatgctgtgctgtatgacatcctgctgttcttgcctgaagggctgctgtagctgtggctcctgctgtaagtttgacgaggatgactctgaacctgtgctgaagggcgtgaagctgcattacacctaa(SEQ ID NO: 17).
Белковая последовательность WT S (штамм Wuhan S-2P) (от N-конца к C-концу):
MFVFLVLLPLVSSQCVNLTTRTQLPPAYTNSFTRGVYYPDKVFRSSVLHSTQDLFLPFFSNVTWFHAIHVSGTNGTKRFDNPVLPFNDGVYFASTEKSNIIRGWIFGTTLDSKTQSLLIVNNATNVVIKVCEFQFCNDPFLGVYYHKNNKSWMESEFRVYSSANNCTFEYVSQPFLMDLEGKQGNFKNLREFVFKNIDGYFKIYSKHTPINLVRDLPQGFSALEPLVDLPIGINITRFQTLLALHRSYLTPGDSSSGWTAGAAAYYVGYLQPRTFLLKYNENGTITDAVDCALDPLSETKCTLKSFTVEKGIYQTSNFRVQPTESIVRFPNITNLCPFGEVFNATRFASVYAWNRKRISNCVADYSVLYNSASFSTFKCYGVSPTKLNDLCFTNVYADSFVIRGDEVRQIAPGQTGKIADYNYKLPDDFTGCVIAWNSNNLDSKVGGNYNYLYRLFRKSNLKPFERDISTEIYQAGSTPCNGVEGFNCYFPLQSYGFQPTNGVGYQPYRVVVLSFELLHAPATVCGPKKSTNLVKNKCVNFNFNGLTGTGVLTESNKKFLPFQQFGRDIADTTDAVRDPQTLEILDITPCSFGGVSVITPGTNTSNQVAVLYQDVNCTEVPVAIHADQLTPTWRVYSTGSNVFQTRAGCLIGAEHVNNSYECDIPIGAGICASYQTQTNSPRRARSVASQSIIAYTMSLGAENSVAYSNNSIAIPTNFTISVTTEILPVSMTKTSVDCTMYICGDSTECSNLLLQYGSFCTQLNRALTGIAVEQDKNTQEVFAQVKQIYKTPPIKDFGGFNFSQILPDPSKPSKRSFIEDLLFNKVTLADAGFIKQYGDCLGDIAARDLICAQKFNGLTVLPPLLTDEMIAQYTSALLAGTITSGWTFGAGAALQIPFAMQMAYRFNGIGVTQNVLYENQKLIANQFNSAIGKIQDSLSSTASALGKLQDVVN  AAEIRASANLAATKMSECVLGQSKRVDFCGKGYHLMSFPQSAPHGVVFLHVTYVPAQEKNFTTAPAICHDGKAHFPREGVFVSNGTHWFVTQRNFYEPQIITTDNTFVSGNCDVVIGIVNNTVYDPLQPELDSFKEELDKYFKNHTSPDVDLGDISGINASVVNIQKEIDRLNEVAKNLNESLIDLQELGKYEQYIKWPWYIWLGFIAGLIAIVMVTIMLCCMTSCCSCLKGCCSCGSCCKFDEDDSEPVLKGVKLHYT(SEQ ID NO: 18)
AAEIRASANLAATKMSECVLGQSKRVDFCGKGYHLMSFPQSAPHGVVFLHVTYVPAQEKNFTTAPAICHDGKAHFPREGVFVSNGTHWFVTQRNFYEPQIITTDNTFVSGNCDVVIGIVNNTVYDPLQPELDSFKEELDKYFKNHTSPDVDLGDISGINASVVNIQKEIDRLNEVAKNLNESLIDLQELGKYEQYIKWPWYIWLGFIAGLIAIVMVTIMLCCMTSCCSCLKGCCSCGSCCKFDEDDSEPVLKGVKLHYT(SEQ ID NO: 18)
Последовательность ДНК или РНК WT S (штамм дельта S-2P) (от 5'-конца к 3'-концу):
atgttcgtcttcctggtcctgctgcctctggtctcctcacagtgcgtcaatctgcgaactcggactcagctgccacctgcttatactaatagcttcaccagaggcgtgtactatcctgacaaggtgtttagaagctccgtgctgcactctacacaggatctgtttctgccattctttagcaacgtgacctggttccacgccatccacgtgagcggcaccaatggcacaaagcggttcgacaatcccgtgctgccttttaacgatggcgtgtacttcgcctctatcgagaagagcaacatcatcagaggctggatctttggcaccacactggactccaagacacagtctctgctgatcgtgaacaatgccaccaacgtggtcatcaaggtgtgcgagttccagttttgtaatgatcccttcctggacgtgtactatcacaagaacaataagagctggatggagtccggagtgtattctagcgccaacaactgcacatttgagtacgtgagccagcctttcctgatggacctggagggcaagcagggcaatttcaagaacctgagggagttcgtgtttaagaatatcgacggctacttcaaaatctactctaagcacacccccatcaacctggtgcgcgacctgcctcagggcttcagcgccctggagcccctggtggatctgcctatcggcatcaacatcacccggtttcagacactgctggccctgcacagaagctacctgacacccggcgactcctctagcggatggaccgccggcgctgccgcctactatgtgggctacctccagccccggaccttcctgctgaagtacaacgagaatggcaccatcacagacgcagtggattgcgccctggaccccctgagcgagacaaagtgtacactgaagtcctttaccgtggagaagggcatctatcagacatccaatttcagggtgcagccaaccgagtctatcgtgcgctttcctaatatcacaaacctgtgcccatttggcgaggtgttcaacgcaacccgcttcgccagcgtgtacgcctggaataggaagcggatcagcaactgcgtggccgactatagcgtgctgtacaactccgcctctttcagcacctttaagtgctatggcgtgtcccccacaaagctgaatgacctgtgctttaccaacgtctacgccgattctttcgtgatcaggggcgacgaggtgcgccagatcgcccccggccagacaggcaagatcgcagactacaattataagctgccagacgatttcaccggctgcgtgatcgcctggaacagcaacaatctggattccaaagtgggcggcaactacaattatcggtaccggctgtttagaaagagcaatctgaagcccttcgagagggacatctctacagaaatctaccaggccggcagcaagccttgcaatggcgtggagggctttaactgttatttcccactccagtcctacggcttccagcccacaaacggcgtgggctatcagccttaccgcgtggtggtgctgagctttgagctgctgcacgccccagcaacagtgtgcggccccaagaagtccaccaatctggtgaagaacaagtgcgtgaacttcaacttcaacggcctgaccggcacaggcgtgctgaccgagtccaacaagaagttcctgccatttcagcagttcggcagggacatcgcagataccacagacgccgtgcgcgacccacagaccctggagatcctggacatcacaccctgctctttcggcggcgtgagcgtgatcacacccggcaccaatacaagcaaccaggtggccgtgctgtatcagggcgtgaattgtaccgaggtgcccgtggctatccacgccgatcagctgaccccaacatggcgggtgtacagcaccggctccaacgtcttccagacaagagccggatgcctgatcggagcagagcacgtgaacaattcctatgagtgcgacatcccaatcggcgccggcatctgtgcctcttaccagacccagacaaactctcgcagaagagcccggagcgtggcctcccagtctatcatcgcctataccatgtccctgggcgccgagaacagcgtggcctactctaacaatagcatcgccatcccaaccaacttcacaatctctgtgaccacagagatcctgcccgtgtccatgaccaagacatctgtggactgcacaatgtatatctgtggcgattctaccgagtgcagcaacctgctgctccagtacggcagcttttgtacccagctgaatagagccctgacaggcatcgccgtggagcaggataagaacacacaggaggtgttcgcccaggtgaagcaaatctacaagaccccccctatcaaggactttggcggcttcaatttttcccagatcctgcctgatccatccaagccttctaagcggagctttatcgaggacctgctgttcaacaaggtgaccctggccgatgccggcttcatcaagcagtatggcgattgcctgggcgacatcgcagccagggacctgatctgcgcccagaagtttaatggcctgaccgtgctgccacccctgctgacagatgagatgatcgcacagtacacaagcgccctgctggccggcaccatcacatccggatggaccttcggcgcaggagccgccctccagatcccctttgccatgcagatggcctataggttcaacggcatcggcgtgacccagaatgtgctgtacgagaaccagaagctgatcgccaatcagtttaactccgccatcggcaagatccaggacagcctgtcctctacagccagcgccctgggcaagctccagaatgtggtgaatcagaacgcccaggccctgaataccctggtgaagcagctgagcagcaacttcggcgccatctctagcgtg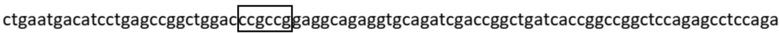
cctatgtgacacagcagctgatcagggccgccgagatcagggccagcgccaatctggcagcaaccaagatgtccgagtgcgtgctgggccagtctaagagagtggacttttgtggcaagggctatcacctgatgtccttccctcagtctgccccacacggcgtggtgtttctgcacgtgacctacgtgcccgcccaggagaagaacttcaccacagcccctgccatctgccacgatggcaaggcccactttccaagggagggcgtgttcgtgtccaacggcacccactggtttgtgacacagcgcaatttctacgagccccagatcatcaccacagacaacaccttcgtgagcggcaactgtgacgtggtcatcggcatcgtgaacaataccgtgtatgatccactccagcccgagctggacagctttaaggaggagctggataagtatttcaagaatcacacctcccctgacgtggatctgggcgacatcagcggcatcaatgcctccgtggtgaacatccagaaggagatcgaccgcctgaacgaggtggctaagaatctgaacgagagcctgatcgacctccaggagctgggcaagtatgagcagtacatcaagtggccctggtacatctggctgggcttcatcgccggcctgatcgccatcgtgatggtgaccatcatgctgtgctgtatgacatcctgctgttcttgcctgaagggctgctgtagctgtggctcctgctgtaagtttgacgaggatgactctgaacctgtgctgaagggcgtgaagctgcattacacctaa(SEQ ID NO: 19).
Белковая последовательность WT S (штамм дельта S-2P) (от N-конца к C-концу):
MFVFLVLLPLVSSQCVNLRTRTQLPPAYTNSFTRGVYYPDKVFRSSVLHSTQDLFLPFFSNVTWFHAIHVSGTNGTKRFDNPVLPFNDGVYFASIEKSNIIRGWIFGTTLDSKTQSLLIVNNATNVVIKVCEFQFCNDPFLDVYYHKNNKSWMESGVYSSANNCTFEYVSQPFLMDLEGKQGNFKNLREFVFKNIDGYFKIYSKHTPINLVRDLPQGFSALEPLVDLPIGINITRFQTLLALHRSYLTPGDSSSGWTAGAAAYYVGYLQPRTFLLKYNENGTITDAVDCALDPLSETKCTLKSFTVEKGIYQTSNFRVQPTESIVRFPNITNLCPFGEVFNATRFASVYAWNRKRISNCVADYSVLYNSASFSTFKCYGVSPTKLNDLCFTNVYADSFVIRGDEVRQIAPGQTGKIADYNYKLPDDFTGCVIAWNSNNLDSKVGGNYNYRYRLFRKSNLKPFERDISTEIYQAGSKPCNGVEGFNCYFPLQSYGFQPTNGVGYQPYRVVVLSFELLHAPATVCGPKKSTNLVKNKCVNFNFNGLTGTGVLTESNKKFLPFQQFGRDIADTTDAVRDPQTLEILDITPCSFGGVSVITPGTNTSNQVAVLYQGVNCTEVPVAIHADQLTPTWRVYSTGSNVFQTRAGCLIGAEHVNNSYECDIPIGAGICASYQTQTNSRRRARSVASQSIIAYTMSLGAENSVAYSNNSIAIPTNFTISVTTEILPVSMTKTSVDCTMYICGDSTECSNLLLQYGSFCTQLNRALTGIAVEQDKNTQEVFAQVKQIYKTPPIKDFGGFNFSQILPDPSKPSKRSFIEDLLFNKVTLADAGFIKQYGDCLGDIAARDLICAQKFNGLTVLPPLLTDEMIAQYTSALLAGTITSGWTFGAGAALQIPFAMQMAYRFNGIGVTQNVLYENQKLIANQFNSAIGKIQDSLSSTASALGKLQNVVNQN

EIRASANLAATKMSECVLGQSKRVDFCGKGYHLMSFPQSAPHGVVFLHVTYVPAQEKNFTTAPAICHDGKAHFPREGVFVSNGTHWFVTQRNFYEPQIITTDNTFVSGNCDVVIGIVNNTVYDPLQPELDSFKEELDKYFKNHTSPDVDLGDISGINASVVNIQKEIDRLNEVAKNLNESLIDLQELGKYEQYIKWPWYIWLGFIAGLIAIVMVTIMLCCMTSCCSCLKGCCSCGSCCKFDEDDSEPVLKGVKLHYT (SEQ ID NO: 20).
Последовательность ДНК или РНК S-(deg-RBD) штамма Wuhan (от 5'-конца к 3'-концу):
Atgttcgtcttcctggtcctgctgcctctggtctcctcacagtgcgtcaatctgacaactcggactcagctgccacctgcttatactaatagcttcaccagaggcgtgtactatcctgacaaggtgtttagaagctccgtgctgcactctacacaggatctgtttctgccattctttagcaacgtgacctggttccacgccatccacgtgagcggcaccaatggcacaaagcggttcgacaatcccgtgctgccttttaacgatggcgtgtacttcgcctctaccgagaagagcaacatcatcagaggctggatctttggcaccacactggactccaagacacagtctctgctgatcgtgaacaatgccaccaacgtggtcatcaaggtgtgcgagttccagttttgtaatgatcccttcctgggcgtgtactatcacaagaacaataagagctggatggagtccgagtttagagtgtattctagcgccaacaactgcacatttgagtacgtgagccagcctttcctgatggacctggagggcaagcagggcaatttcaagaacctgagggagttcgtgtttaagaatatcgacggctacttcaaaatctactctaagcacacccccatcaacctggtgcgcgacctgcctcagggcttcagcgccctggagcccctggtggatctgcctatcggcatcaacatcacccggtttcagacactgctggccctgcacagaagctacctgacacccggcgactcctctagcggatggaccgccggcgctgccgcctactatgtgggctacctccagccccggaccttcctgctgaagtacaacgagaatggcaccatcacagacgcagtggattgcgccctggaccccctgagcgagacaaagtgtacactgaagtcctttac
gctgtacaactccgcctctttcagcacctttaagtgctatggcgtgtcccccacaaagctgaatgacctgtgctttaccaacgtctacgccgattctttcgtgatcaggggcgacgaggtgcgccagatcgcccccggccagacaggcaagatcgcagactacaattataagctgccagacgatttcaccggctgcgtgatcgcctggaacagcaacaatctggattccaaagtgggcggcaactacaattatctgtaccggctgtttagaaagagcaatctgaagcccttcgagagggacatctctacagaaatctaccaggccggcagcaccccttgcaatggcgtggagggctttaactgttatttcccactccagtcctacggcttccagcccacaaacggcgtgggctatcagccttaccgcgtggtggtgctgagctttgagctgctgcacgccccagcaacagtgtgcggccccaagaagtccaccaatctggtgaagaacaagtgcgtgaacttcaacttcaacggcctgaccggcacaggcgtgctgaccgagtccaacaagaagttcctgccatttcagcagttcggcagggacatcgcagataccacagacgccgtgcgcgacccacagaccctggagatcctggacatcacaccctgctctttcggcggcgtgagcgtgatcacacccggcaccaatacaagcaaccaggtggccgtgctgtatcaggacgtgaattgtaccgaggtgcccgtggctatccacgccgatcagctgaccccaacatggcgggtgtacagcaccggctccaacgtcttccagacaagagccggatgcctgatcggagcagagcacgtgaacaattcctatgagtgcgacatcccaatcggcgccggcatctgtgcctcttaccagacccagacaaactctcccagaagagcccggagcgtggcctcccagtctatcatcgcctataccatgtccctgggcgccgagaacagcgtggcctactctaacaatagcatcgccatcccaaccaacttcacaatctctgtgaccacagagatcctgcccgtgtccatgaccaagacatctgtggactgcacaatgtatatctgtggcgattctaccgagtgcagcaacctgctgctccagtacggcagcttttgtacccagctgaatagagccctgacaggcatcgccgtggagcaggataagaacacacaggaggtgttcgcccaggtgaagcaaatctacaagaccccccctatcaaggactttggcggcttcaatttttcccagatcctgcctgatccatccaagccttctaagcggagctttatcgaggacctgctgttcaacaaggtgaccctggccgatgccggcttcatcaagcagtatggcgattgcctgggcgacatcgcagccagggacctgatctgcgcccagaagtttaatggcctgaccgtgctgccacccctgctgacagatgagatgatcgcacagtacacaagcgccctgctggccggcaccatcacatccggatggaccttcggcgcaggagccgccctccagatcccctttgccatgcagatggcctataggttcaacggcatcggcgtgacccagaatgtgctgtacgagaaccagaagctgatcgccaatcagtttaactccgccatcggcaagatccaggacagcctgtcctctacagccagcgccctgggcaagctccaggatgtggtgaatcagaacgcccaggccctgaataccctggtgaagcagctgagcagcaacttcggcgccatctctagcgtgctgaatgacatcctgagccggctggacaaggtggaggcagaggtgcagatcgaccggctgatcaccggccggctccagagcctccagacctatgtgacacagcagctgatcagggccgccgagatcagggccagcgccaatctggcagcaaccaagatgtccgagtgcgtgctgggccagtctaagagagtggacttttgtggcaagggctatcacctgatgtccttccctcagtctgccccacacggcgtggtgtttctgcacgtgacctacgtgcccgcccaggagaagaacttcaccacagcccctgccatctgccacgatggcaaggcccactttccaagggagggcgtgttcgtgtccaacggcacccactggtttgtgacacagcgcaatttctacgagccccagatcatcaccacagacaacaccttcgtgagcggcaactgtgacgtggtcatcggcatcgtgaacaataccgtgtatgatccactccagcccgagctggacagctttaaggaggagctggataagtatttcaagaatcacacctcccctgacgtggatctgggcgacatcagcggcatcaatgcctccgtggtgaacatccagaaggagatcgaccgcctgaacgaggtggctaagaatctgaacgagagcctgatcgacctccaggagctgggcaagtatgagcagtacatcaagtggccctggtacatctggctgggcttcatcgccggcctgatcgccatcgtgatggtgaccatcatgctgtgctgtatgacatcctgctgttcttgcctgaagggctgctgtagctgtggctcctgctgtaagtttgacgaggatgactctgaacctgtgctgaagggcgtgaagctgcattacacctaa (SEQ ID NO: 3).
Белковая последовательность S-(deg-RBD) для штамма Wuhan (от N-конца к C-концу):
MFVFLVLLPLVSSQCVNLTTRTQLPPAYTNSFTRGVYYPDKVFRSSVLHSTQDLFLPFFSNVTWFHAIHVSGTNGTKRFDNPVLPFNDGVYFASTEKSNIIRGWIFGTTLDSKTQSLLIVNNATNVVIKVCEFQFCNDPFLGVYYHKNNKSWMESEFRVYSSANNCTFEYVSQPFLMDLEGKQGNFKNLREFVFKNIDGYFKIYSKHTPINLVRDLPQGFSALEPLVDLPIGINITRFQTLLALHRSYLTPGDSSSGWTAGAAAYYVGYLQPRTFLLKYNENGTITDAVDCALD

RKRISNCVADYSVLYNSASFSTFKCYGVSPTKLNDLCFTNVYADSFVIRGDEVRQIAPGQTGKIADYNYKLPDDFTGCVIAWNSNNLDSKVGGNYNYLYRLFRKSNLKPFERDISTEIYQAGSTPCNGVEGFNCYFPLQSYGFQPTNGVGYQPYRVVVLSFELLHAPATVCGPKKSTNLVKNKCVNFNFNGLTGTGVLTESNKKFLPFQQFGRDIADTTDAVRDPQTLEILDITPCSFGGVSVITPGTNTSNQVAVLYQDVNCTEVPVAIHADQLTPTWRVYSTGSNVFQTRAGCLIGAEHVNNSYECDIPIGAGICASYQTQTNSPRRARSVASQSIIAYTMSLGAENSVAYSNNSIAIPTNFTISVTTEILPVSMTKTSVDCTMYICGDSTECSNLLLQYGSFCTQLNRALTGIAVEQDKNTQEVFAQVKQIYKTPPIKDFGGFNFSQILPDPSKPSKRSFIEDLLFNKVTLADAGFIKQYGDCLGDIAARDLICAQKFNGLTVLPPLLTDEMIAQYTSALLAGTITSGWTFGAGAALQIPFAMQMAYRFNGIGVTQNVLYENQKLIANQFNSAIGKIQDSLSSTASALGKLQDVVNQNAQALNTLVKQLSSNFGAISSVLNDILSRLDPPEAEVQIDRLITGRLQSLQTYVTQQLIRAAEIRASANLAATKMSECVLGQSKRVDFCGKGYHLMSFPQSAPHGVVFLHVTYVPAQEKNFTTAPAICHDGKAHFPREGVFVSNGTHWFVTQRNFYEPQIITTDNTFVSGNCDVVIGIVNNTVYDPLQPELDSFKEELDKYFKNHTSPDVDLGDISGINASVVNIQKEIDRLNEVAKNLNESLIDLQELGKYEQYIKWPWYIWLGFIAGLIAIVMVTIMLCCMTSCCSCLKGCCSCGSCCKFDEDDSEPVLKGVKLHYT (SEQ ID NO: 4).
Последовательность ДНК или РНК S-(deg-RBD) штамма дельта (от 5'-конца к 3'-концу):
atgttcgtcttcctggtcctgctgcctctggtctcctcacagtgcgtcaatctgcgaactcggactcagctgccacctgcttatactaatagcttcaccagaggcgtgtactatcctgacaaggtgtttagaagctccgtgctgcactctacacaggatctgtttctgccattctttagcaacgtgacctggttccacgccatccacgtgagcggcaccaatggcacaaagcggttcgacaatcccgtgctgccttttaacgatggcgtgtacttcgcctctatcgagaagagcaacatcatcagaggctggatctttggcaccacactggactccaagacacagtctctgctgatcgtgaacaatgccaccaacgtggtcatcaaggtgtgcgagttccagttttgtaatgatcccttcctggacgtgtactatcacaagaacaataagagctggatggagtccggagtgtattctagcgccaacaactgcacatttgagtacgtgagccagcctttcctgatggacctggagggcaagcagggcaatttcaagaacctgagggagttcgtgtttaagaatatcgacggctacttcaaaatctactctaagcacacccccatcaacctggtgcgcgacctgcctcagggcttcagcgccctggagcccctggtggatctgcctatcggcatcaacatcacccggtttcagacactgctggccctgcacagaagctacctgacacccggcgactcctctagcggatggaccgccggcgctgccgcctactatgtgggctacctccagccccggaccttcctgctgaagtacaacgagaatggcaccatcacagacgcagtggattgcgccctggaccccctgagcgagacaaagtgtacactgaagtcctttaccgtggagaagggcatctatcagacatccaatttcagggtgcagccagccgaggcgatcgtgcgctttcctgaaatcacaaacctgtgcccatttggcgaggtgttcgaggcaacccgcttcgccagcgtgtacgcctggaataggaagcggatcagcaactgcgtggccgactatagcgtgctgtacaactccgcctctttcagcacctttaagtgctatggcgtgtcccccacaaagctgaatgacctgtgctttaccaacgtctacgccgattctttcgtgatcaggggcgacgaggtgcgccagatcgcccccggccagacaggcaagatcgcagactacaattataagctgccagacgatttcaccggctgcgtgatcgcctggaacagcaacaatctggattccaaagtgggcggcaactacaattatcggtaccggctgtttagaaagagcaatctgaagcccttcgagagggacatctctacagaaatctaccaggccggcagcaagccttgcaatggcgtggagggctttaactgttatttcccactccagtcctacggcttccagcccacaaacggcgtgggctatcagccttaccgcgtggtggtgctgagctttgagctgctgcacgccccagcaacagtgtgcggccccaagaagtccaccaatctggtgaagaacaagtgcgtgaacttcaacttcaacggcctgaccggcacaggcgtgctgaccgagtccaacaagaagttcctgccatttcagcagttcggcagggacatcgcagataccacagacgccgtgcgcgacccacagaccctggagatcctggacatcacaccctgctctttcggcggcgtgagcgtgatcacacccggcaccaatacaagcaaccaggtggccgtgctgtatcagggcgtgaattgtaccgaggtgcccgtggctatccacgccgatcagctgaccccaacatggcgggtgtacagcaccggctccaacgtcttccagacaagagccggatgcctgatcggagcagagcacgtgaacaattcctatgagtgcgacatcccaatcggcgccggcatctgtgcctcttaccagacccagacaaactctcgcagaagagcccggagcgtggcctcccagtctatcatcgcctataccatgtccctgggcgccgagaacagcgtggcctactctaacaatagcatcgccatcccaaccaacttcacaatctctgtgaccacagagatcctgcccgtgtccatgaccaagacatctgtggactgcacaatgtatatctgtggcgattctaccgagtgcagcaacctgctgctccagtacggcagcttttgtacccagctgaatagagccctgacaggcatcgccgtggagcaggataagaacacacaggaggtgttcgcccaggtgaagcaaatctacaagaccccccctatcaaggactttggcggcttcaatttttcccagatcctgcctgatccatccaagccttctaagcggagctttatcgaggacctgctgttcaacaaggtgaccctggccgatgccggcttcatcaagcagtatggcgattgcctgggcgacatcgcagccagggacctgatctgcgcccagaagtttaatggcctgaccgtgctgccacccctgctgacagatgagatgatcgcacagtacacaagcgccctgctggccggcaccatcacatccggatggaccttcggcgcaggagccgccctccagatcccctttgccatgcagatggcctataggttcaacggcatcggcgtgacccagaatgtgctgtacgagaaccagaagctgatcgccaatcagtttaactccgccatcggcaagatccaggacagcctgtcctctacagccagcgccctgggcaagctccagaatgtggtgaatcagaacgcccaggccctgaataccctggtgaagcagctgagcagcaacttcggcgccatctctagcgtgctgaatgacatcctgagccggctggacaaggtggaggcagaggtgcagatcgaccggctgatcaccggccggctccagagcctccagacctatgtgacacagcagctgatcagggccgccgagatcagggccagcgccaatctggcagcaaccaagatgtccgagtgcgtgctgggccagtctaagagagtggacttttgtggcaagggctatcacctgatgtccttccctcagtctgccccacacggcgtggtgtttctgcacgtgacctacgtgcccgcccaggagaagaacttcaccacagcccctgccatctgccacgatggcaaggcccactttccaagggagggcgtgttcgtgtccaacggcacccactggtttgtgacacagcgcaatttctacgagccccagatcatcaccacagacaacaccttcgtgagcggcaactgtgacgtggtcatcggcatcgtgaacaataccgtgtatgatccactccagcccgagctggacagctttaaggaggagctggataagtatttcaagaatcacacctcccctgacgtggatctgggcgacatcagcggcatcaatgcctccgtggtgaacatccagaaggagatcgaccgcctgaacgaggtggctaagaatctgaacgagagcctgatcgacctccaggagctgggcaagtatgagcagtacatcaagtggccctggtacatctggctgggcttcatcgccggcctgatcgccatcgtgatggtgaccatcatgctgtgctgtatgacatcctgctgttcttgcctgaagggctgctgtagctgtggctcctgctgtaagtttgacgaggatgactctgaacctgtgctgaagggcgtgaagctgcattacacctaa (SEQ ID NO: 21)
Белковая последовательность S-(deg-RBD) для штамма дельта (от N-конца к C-концу):
MFVFLVLLPLVSSQCVNLRTRTQLPPAYTNSFTRGVYYPDKVFRSSVLHSTQDLFLPFFSNVTWFHAIHVSGTNGTKRFDNPVLPFNDGVYFASIEKSNIIRGWIFGTTLDSKTQSLLIVNNATNVVIKVCEFQFCNDPFLDVYYHKNNKSWMESGVYSSANNCTFEYVSQPFLMDLEGKQGNFKNLREFVFKNIDGYFKIYSKHTPINLVRDLPQGFSALEPLVDLPIGINITRFQTLLALHRSYLTPGDSSSGWTAGAAAYYVGYLQPRTFLLKYNENGTITDAVDCALDPLSETKCTLKSFTVEKGIYQTSNFRVQPAEAIVRFPQITNLCPFGEVFQATRFASVYAWNRKRISNCVADYSVLYNSASFSTFKCYGVSPTKLNDLCFTNVYADSFVIRGDEVRQIAPGQTGKIADYNYKLPDDFTGCVIAWNSNNLDSKVGGNYNYRYRLFRKSNLKPFERDISTEIYQAGSKPCNGVEGFNCYFPLQSYGFQPTNGVGYQPYRVVVLSFELLHAPATVCGPKKSTNLVKNKCVNFNFNGLTGTGVLTESNKKFLPFQQFGRDIADTTDAVRDPQTLEILDITPCSFGGVSVITPGTNTSNQVAVLYQGVNCTEVPVAIHADQLTPTWRVYSTGSNVFQTRAGCLIGAEHVNNSYECDIPIGAGICASYQTQTNSRRRARSVASQSIIAYTMSLGAENSVAYSNNSIAIPTNFTISVTTEILPVSMTKTSVDCTMYICGDSTECSNLLLQYGSFCTQLNRALTGIAVEQDKNTQEVFAQVKQIYKTPPIKDFGGFNFSQILPDPSKPSKRSFIEDLLFNKVTLADAGFIKQYGDCLGDIAARDLICAQKFNGLTVLPPLLTDEMIAQYTSALLAGTITSGWTFGAGAALQIPFAMQMAYRFNGIGVTQNVLYENQKLIANQFNSAIGKIQDSLSSTASALGKLQNVVNQNAQALNTLVKQLSSNFGAISSVLNDILSRLDKVEAEVQIDRLITGRLQSLQTYVTQQLIRAAEIRASANLAATKMSECVLGQSKRVDFCGKGYHLMSFPQSAPHGVVFLHVTYVPAQEKNFTTAPAICHDGKAHFPREGVFVSNGTHWFVTQRNFYEPQIITTDNTFVSGNCDVVIGIVNNTVYDPLQPELDSFKEELDKYFKNHTSPDVDLGDISGINASVVNIQKEIDRLNEVAKNLNESLIDLQELGKYEQYIKWPWYIWLGFIAGLIAIVMVTIMLCCMTSCCSCLKGCCSCGSCCKFDEDDSEPVLKGVKLHYT (SEQ ID NO: 22)
Последовательность ДНК или РНК S-(deg-RBD) штамма Wuhan S-2P (от 5'-конца к 3'-концу):
atgttcgtcttcctggtcctgctgcctctggtctcctcacagtgcgtcaatctgacaactcggactcagctgccacctgcttatactaatagcttcaccagaggcgtgtactatcctgacaaggtgtttagaagctccgtgctgcactctacacaggatctgtttctgccattctttagcaacgtgacctggttccacgccatccacgtgagcggcaccaatggcacaaagcggttcgacaatcccgtgctgccttttaacgatggcgtgtacttcgcctctaccgagaagagcaacatcatcagaggctggatctttggcaccacactggactccaagacacagtctctgctgatcgtgaacaatgccaccaacgtggtcatcaaggtgtgcgagttccagttttgtaatgatcccttcctgggcgtgtactatcacaagaacaataagagctggatggagtccgagtttagagtgtattctagcgccaacaactgcacatttgagtacgtgagccagcctttcctgatggacctggagggcaagcagggcaatttcaagaacctgagggagttcgtgtttaagaatatcgacggctacttcaaaatctactctaagcacacccccatcaacctggtgcgcgacctgcctcagggcttcagcgccctggagcccctggtggatctgcctatcggcatcaacatcacccggtttcagacactgctggccctgcacagaagctacctgacacccggcgactcctctagcggatggaccgccggcgctgccgcctactatgtgggctacctccagccccggaccttcctgctgaagtacaacgagaatggcaccatcacagacgcagtggattgcgccctggaccccctgagcgagacaaagtgtacactgaagtcctttaccgtggagaagggcatctatcagacatccaatttcagggtgcagccagccgaggcgatcgtgcgctttcctgaaatcacaaacctgtgcccatttggcgaggtgttcgaggcaacccgcttcgccagcgtgtacgcctggaataggaagcggatcagcaactgcgtggccgactatagcgtgctgtacaactccgcctctttcagcacctttaagtgctatggcgtgtcccccacaaagctgaatgacctgtgctttaccaacgtctacgccgattctttcgtgatcaggggcgacgaggtgcgccagatcgcccccggccagacaggcaagatcgcagactacaattataagctgccagacgatttcaccggctgcgtgatcgcctggaacagcaacaatctggattccaaagtgggcggcaactacaattatctgtaccggctgtttagaaagagcaatctgaagcccttcgagagggacatctctacagaaatctaccaggccggcagcaccccttgcaatggcgtggagggctttaactgttatttcccactccagtcctacggcttccagcccacaaacggcgtgggctatcagccttaccgcgtggtggtgctgagctttgagctgctgcacgccccagcaacagtgtgcggccccaagaagtccaccaatctggtgaagaacaagtgcgtgaacttcaacttcaacggcctgaccggcacaggcgtgctgaccgagtccaacaagaagttcctgccatttcagcagttcggcagggacatcgcagataccacagacgccgtgcgcgacccacagaccctggagatcctggacatcacaccctgctctttcggcggcgtgagcgtgatcacacccggcaccaatacaagcaaccaggtggccgtgctgtatcaggacgtgaattgtaccgaggtgcccgtggctatccacgccgatcagctgaccccaacatggcgggtgtacagcaccggctccaacgtcttccagacaagagccggatgcctgatcggagcagagcacgtgaacaattcctatgagtgcgacatcccaatcggcgccggcatctgtgcctcttaccagacccagacaaactctcccagaagagcccggagcgtggcctcccagtctatcatcgcctataccatgtccctgggcgccgagaacagcgtggcctactctaacaatagcatcgccatcccaaccaacttcacaatctctgtgaccacagagatcctgcccgtgtccatgaccaagacatctgtggactgcacaatgtatatctgtggcgattctaccgagtgcagcaacctgctgctccagtacggcagcttttgtacccagctgaatagagccctgacaggcatcgccgtggagcaggataagaacacacaggaggtgttcgcccaggtgaagcaaatctacaagaccccccctatcaaggactttggcggcttcaatttttcccagatcctgcctgatccatccaagccttctaagcggagctttatcgaggacctgctgttcaacaaggtgaccctggccgatgccggcttcatcaagcagtatggcgattgcctgggcgacatcgcagccagggacctgatctgcgcccagaagtttaatggcctgaccgtgctgccacccctgctgacagatgagatgatcgcacagtacacaagcgccctgctggccggcaccatcacatccggatggaccttcggcgcaggagccgccctccagatcccctttgccatgcagatggcctataggttcaacggcatcggcgtgacccagaatgtgctgtacgagaaccagaagctgatcgccaatcagtttaactccgccatcggcaagatccaggacagcctgtcctctacagccagcgccctgggcaagctccaggatgtggtgaatcagaacgcccaggccctgaataccctggtgaagcagctgagcagcaacttcggcgccatctct
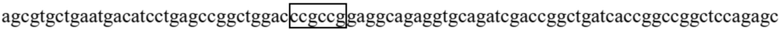
ctccagacctatgtgacacagcagctgatcagggccgccgagatcagggccagcgccaatctggcagcaaccaagatgtccgagtgcgtgctgggccagtctaagagagtggacttttgtggcaagggctatcacctgatgtccttccctcagtctgccccacacggcgtggtgtttctgcacgtgacctacgtgcccgcccaggagaagaacttcaccacagcccctgccatctgccacgatggcaaggcccactttccaagggagggcgtgttcgtgtccaacggcacccactggtttgtgacacagcgcaatttctacgagccccagatcatcaccacagacaacaccttcgtgagcggcaactgtgacgtggtcatcggcatcgtgaacaataccgtgtatgatccactccagcccgagctggacagctttaaggaggagctggataagtatttcaagaatcacacctcccctgacgtggatctgggcgacatcagcggcatcaatgcctccgtggtgaacatccagaaggagatcgaccgcctgaacgaggtggctaagaatctgaacgagagcctgatcgacctccaggagctgggcaagtatgagcagtacatcaagtggccctggtacatctggctgggcttcatcgccggcctgatcgccatcgtgatggtgaccatcatgctgtgctgtatgacatcctgctgttcttgcctgaagggctgctgtagctgtggctcctgctgtaagtttgacgaggatgactctgaacctgtgctgaagggcgtgaagctgcattacacctaa(SEQ ID NO: 23)
Белковая последовательность S-(deg-RBD) для штамма Wuhan S-2P (от N-конца к C-концу):
MFVFLVLLPLVSSQCVNLTTRTQLPPAYTNSFTRGVYYPDKVFRSSVLHSTQDLFLPFFSNVTWFHAIHVSGTNGTKRFDNPVLPFNDGVYFASTEKSNIIRGWIFGTTLDSKTQSLLIVNNATNVVIKVCEFQFCNDPFLGVYYHKNNKSWMESEFRVYSSANNCTFEYVSQPFLMDLEGKQGNFKNLREFVFKNIDGYFKIYSKHTPINLVRDLPQGFSALEPLVDLPIGINITRFQTLLALHRSYLTPGDSSSGWTAGAAAYYVGYLQPRTFLLKYNENGTITDAVDCALDPLSETKCTLKSFTVEKGIYQTSNFRVQPAEAIVRFPQITNLCPFGEVFQATRFASVYAWNRKRISNCVADYSVLYNSASFSTFKCYGVSPTKLNDLCFTNVYADSFVIRGDEVRQIAPGQTGKIADYNYKLPDDFTGCVIAWNSNNLDSKVGGNYNYLYRLFRKSNLKPFERDISTEIYQAGSTPCNGVEGFNCYFPLQSYGFQPTNGVGYQPYRVVVLSFELLHAPATVCGPKKSTNLVKNKCVNFNFNGLTGTGVLTESNKKFLPFQQFGRDIADTTDAVRDPQTLEILDITPCSFGGVSVITPGTNTSNQVAVLYQDVNCTEVPVAIHADQLTPTWRVYSTGSNVFQTRAGCLIGAEHVNNSYECDIPIGAGICASYQTQTNSPRRARSVASQSIIAYTMSLGAENSVAYSNNSIAIPTNFTISVTTEILPVSMTKTSVDCTMYICGDSTECSNLLLQYGSFCTQLNRALTGIAVEQDKNTQEVFAQVKQIYKTPPIKDFGGFNFSQILPDPSKPSKRSFIEDLLFNKVTLADAGFIKQYGDCLGDIAARDLICAQKFNGLTVLPPLLTDEMIAQYTSALLAGTITSGWTFGAGAALQIPFAMQMAYRFNGIGVTQNVLYENQKLIANQFNSAIGKIQDSLSSTASALGKLQDVV
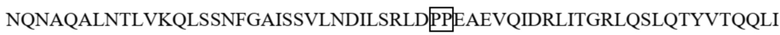
RAAEIRASANLAATKMSECVLGQSKRVDFCGKGYHLMSFPQSAPHGVVFLHVTYVPAQEKNFTTAPAICHDGKAHFPREGVFVSNGTHWFVTQRNFYEPQIITTDNTFVSGNCDVVIGIVNNTVYDPLQPELDSFKEELDKYFKNHTSPDVDLGDISGINASVVNIQKEIDRLNEVAKNLNESLIDLQELGKYEQYIKWPWYIWLGFIAGLIAIVMVTIMLCCMTSCCSCLKGCCSCGSCCKFDEDDSEPVLKGVKLHYT(SEQ ID NO: 24)
Последовательность ДНК или РНК S-(deg-RBD) для штамма дельта S-2P (от 5'-конца к 3'-концу):
atgttcgtcttcctggtcctgctgcctctggtctcctcacagtgcgtcaatctgcgaactcggactcagctgccacctgcttatactaatagcttcaccagaggcgtgtactatcctgacaaggtgtttagaagctccgtgctgcactctacacaggatctgtttctgccattctttagcaacgtgacctggttccacgccatccacgtgagcggcaccaatggcacaaagcggttcgacaatcccgtgctgccttttaacgatggcgtgtacttcgcctctatcgagaagagcaacatcatcagaggctggatctttggcaccacactggactccaagacacagtctctgctgatcgtgaacaatgccaccaacgtggtcatcaaggtgtgcgagttccagttttgtaatgatcccttcctggacgtgtactatcacaagaacaataagagctggatggagtccggagtgtattctagcgccaacaactgcacatttgagtacgtgagccagcctttcctgatggacctggagggcaagcagggcaatttcaagaacctgagggagttcgtgtttaagaatatcgacggctacttcaaaatctactctaagcacacccccatcaacctggtgcgcgacctgcctcagggcttcagcgccctggagcccctggtggatctgcctatcggcatcaacatcacccggtttcagacactgctggccctgcacagaagctacctgacacccggcgactcctctagcggatggaccgccggcgctgccgcctactatgtgggctacctccagccccggaccttcctgctgaagtacaacgagaatggcaccatcacagacgcagtggattgcgccctggaccccctgagcgagacaaagtgtacactgaagtcctttaccgtggagaagggcatctatcagacatccaatttcagggtgcagccagccgaggcgatcgtgcgctttcctgaaatcacaaacctgtgcccatttggcgaggtgttcgaggcaacccgcttcgccagcgtgtacgcctggaataggaagcggatcagcaactgcgtggccgactatagcgtgctgtacaactccgcctctttcagcacctttaagtgctatggcgtgtcccccacaaagctgaatgacctgtgctttaccaacgtctacgccgattctttcgtgatcaggggcgacgaggtgcgccagatcgcccccggccagacaggcaagatcgcagactacaattataagctgccagacgatttcaccggctgcgtgatcgcctggaacagcaacaatctggattccaaagtgggcggcaactacaattatcggtaccggctgtttagaaagagcaatctgaagcccttcgagagggacatctctacagaaatctaccaggccggcagcaagccttgcaatggcgtggagggctttaactgttatttcccactccagtcctacggcttccagcccacaaacggcgtgggctatcagccttaccgcgtggtggtgctgagctttgagctgctgcacgccccagcaacagtgtgcggccccaagaagtccaccaatctggtgaagaacaagtgcgtgaacttcaacttcaacggcctgaccggcacaggcgtgctgaccgagtccaacaagaagttcctgccatttcagcagttcggcagggacatcgcagataccacagacgccgtgcgcgacccacagaccctggagatcctggacatcacaccctgctctttcggcggcgtgagcgtgatcacacccggcaccaatacaagcaaccaggtggccgtgctgtatcagggcgtgaattgtaccgaggtgcccgtggctatccacgccgatcagctgaccccaacatggcgggtgtacagcaccggctccaacgtcttccagacaagagccggatgcctgatcggagcagagcacgtgaacaattcctatgagtgcgacatcccaatcggcgccggcatctgtgcctcttaccagacccagacaaactctcgcagaagagcccggagcgtggcctcccagtctatcatcgcctataccatgtccctgggcgccgagaacagcgtggcctactctaacaatagcatcgccatcccaaccaacttcacaatctctgtgaccacagagatcctgcccgtgtccatgaccaagacatctgtggactgcacaatgtatatctgtggcgattctaccgagtgcagcaacctgctgctccagtacggcagcttttgtacccagctgaatagagccctgacaggcatcgccgtggagcaggataagaacacacaggaggtgttcgcccaggtgaagcaaatctacaagaccccccctatcaaggactttggcggcttcaatttttcccagatcctgcctgatccatccaagccttctaagcggagctttatcgaggacctgctgttcaacaaggtgaccctggccgatgccggcttcatcaagcagtatggcgattgcctgggcgacatcgcagccagggacctgatctgcgcccagaagtttaatggcctgaccgtgctgccacccctgctgacagatgagatgatcgcacagtacacaagcgccctgctggccggcaccatcacatccggatggaccttcggcgcaggagccgccctccagatcccctttgccatgcagatggcctataggttcaacggcatcggcgtgacccagaatgtgctgtacgagaaccagaagctgatcgccaatcagtttaactccgccatcggcaagatccaggacagcctgtcctctacagccagcgccctgggcaagctccagaatgtggtgaatcagaacgcccaggccctgaataccctggtgaagcagctgagcagcaacttcggcgccatctct
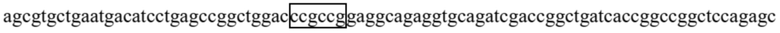
ctccagacctatgtgacacagcagctgatcagggccgccgagatcagggccagcgccaatctggcagcaaccaagatgtccgagtgcgtgctgggccagtctaagagagtggacttttgtggcaagggctatcacctgatgtccttccctcagtctgccccacacggcgtggtgtttctgcacgtgacctacgtgcccgcccaggagaagaacttcaccacagcccctgccatctgccacgatggcaaggcccactttccaagggagggcgtgttcgtgtccaacggcacccactggtttgtgacacagcgcaatttctacgagccccagatcatcaccacagacaacaccttcgtgagcggcaactgtgacgtggtcatcggcatcgtgaacaataccgtgtatgatccactccagcccgagctggacagctttaaggaggagctggataagtatttcaagaatcacacctcccctgacgtggatctgggcgacatcagcggcatcaatgcctccgtggtgaacatccagaaggagatcgaccgcctgaacgaggtggctaagaatctgaacgagagcctgatcgacctccaggagctgggcaagtatgagcagtacatcaagtggccctggtacatctggctgggcttcatcgccggcctgatcgccatcgtgatggtgaccatcatgctgtgctgtatgacatcctgctgttcttgcctgaagggctgctgtagctgtggctcctgctgtaagtttgacgaggatgactctgaacctgtgctgaagggcgtgaagctgcattacacctaa(SEQ ID NO: 25)
Белковая последовательность S-(deg-RBD) для штамма дельта S-2P (от N-конца к C-концу):
MFVFLVLLPLVSSQCVNLRTRTQLPPAYTNSFTRGVYYPDKVFRSSVLHSTQDLFLPFFSNVTWFHAIHVSGTNGTKRFDNPVLPFNDGVYFASIEKSNIIRGWIFGTTLDSKTQSLLIVNNATNVVIKVCEFQFCNDPFLDVYYHKNNKSWMESGVYSSANNCTFEYVSQPFLMDLEGKQGNFKNLREFVFKNIDGYFKIYSKHTPINLVRDLPQGFSALEPLVDLPIGINITRFQTLLALHRSYLTPGDSSSGWTAGAAAYYVGYLQPRTFLLKYNENGTITDAVDCALDPLSETKCTLKSFTVEKGIYQTSNFRVQPAEAIVRFPQITNLCPFGEVFQATRFASVYAWNRKRISNCVADYSVLYNSASFSTFKCYGVSPTKLNDLCFTNVYADSFVIRGDEVRQIAPGQTGKIADYNYKLPDDFTGCVIAWNSNNLDSKVGGNYNYRYRLFRKSNLKPFERDISTEIYQAGSKPCNGVEGFNCYFPLQSYGFQPTNGVGYQPYRVVVLSFELLHAPATVCGPKKSTNLVKNKCVNFNFNGLTGTGVLTESNKKFLPFQQFGRDIADTTDAVRDPQTLEILDITPCSFGGVSVITPGTNTSNQVAVLYQGVNCTEVPVAIHADQLTPTWRVYSTGSNVFQTRAGCLIGAEHVNNSYECDIPIGAGICASYQTQTNSRRRARSVASQSIIAYTMSLGAENSVAYSNNSIAIPTNFTISVTTEILPVSMTKTSVDCTMYICGDSTECSNLLLQYGSFCTQLNRALTGIAVEQDKNTQEVFAQVKQIYKTPPIKDFGGFNFSQILPDPSKPSKRSFIEDLLFNKVTLADAGFIKQYGDCLGDIAARDLICAQKFNGLTVLPPLLTDEMIAQYTSALLAGTITSGWTFGAGAALQIPFAMQMAYRFNGIGVTQNVLYENQKLIANQFNSAIGKIQDSLSSTASALGKLQNVVN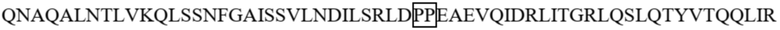 AAEIRASANLAATKMSECVLGQSKRVDFCGKGYHLMSFPQSAPHGVVFLHVTYVPAQEKNFTTAPAICHDGKAHFPREGVFVSNGTHWFVTQRNFYEPQIITTDNTFVSGNCDVVIGIVNNTVYDPLQPELDSFKEELDKYFKNHTSPDVDLGDISGINASVVNIQKEIDRLNEVAKNLNESLIDLQELGKYEQYIKWPWYIWLGFIAGLIAIVMVTIMLCCMTSCCSCLKGCCSCGSCCKFDEDDSEPVLKGVKLHYT (SEQ ID NO: 26)
AAEIRASANLAATKMSECVLGQSKRVDFCGKGYHLMSFPQSAPHGVVFLHVTYVPAQEKNFTTAPAICHDGKAHFPREGVFVSNGTHWFVTQRNFYEPQIITTDNTFVSGNCDVVIGIVNNTVYDPLQPELDSFKEELDKYFKNHTSPDVDLGDISGINASVVNIQKEIDRLNEVAKNLNESLIDLQELGKYEQYIKWPWYIWLGFIAGLIAIVMVTIMLCCMTSCCSCLKGCCSCGSCCKFDEDDSEPVLKGVKLHYT (SEQ ID NO: 26)
Последовательность ДНК или РНК S-(deg-S2) для штамма Wuhan (от 5'-конца к 3'-концу):
atgttcgtcttcctggtcctgctgcctctggtctcctcacagtgcgtcaatctgacaactcggactcagctgccacctgcttatactaatagcttcaccagaggcgtgtactatcctgacaaggtgtttagaagctccgtgctgcactctacacaggatctgtttctgccattctttagcaacgtgacctggttccacgccatccacgtgagcggcaccaatggcacaaagcggttcgacaatcccgtgctgccttttaacgatggcgtgtacttcgcctctaccgagaagagcaacatcatcagaggctggatctttggcaccacactggactccaagacacagtctctgctgatcgtgaacaatgccaccaacgtggtcatcaaggtgtgcgagttccagttttgtaatgatcccttcctgggcgtgtactatcacaagaacaataagagctggatggagtccgagtttagagtgtattctagcgccaacaactgcacatttgagtacgtgagccagcctttcctgatggacctggagggcaagcagggcaatttcaagaacctgagggagttcgtgtttaagaatatcgacggctacttcaaaatctactctaagcacacccccatcaacctggtgcgcgacctgcctcagggcttcagcgccctggagcccctggtggatctgcctatcggcatcaacatcacccggtttcagacactgctggccctgcacagaagctacctgacacccggcgactcctctagcggatggaccgccggcgctgccgcctactatgtgggctacctccagccccggaccttcctgctgaagtacaacgagaatggcaccatcacagacgcagtggattgcgccctggaccccctgagcgagacaaagtgtacactgaagtcctttaccgtggagaagggcatctatcagacatccaatttcagggtgcagccaaccgagtctatcgtgcgctttcctaatatcacaaacctgtgcccatttggcgaggtgttcaacgcaacccgcttcgccagcgtgtacgcctggaataggaagcggatcagcaactgcgtggccgactatagcgtgctgtacaactccgcctctttcagcacctttaagtgctatggcgtgtcccccacaaagctgaatgacctgtgctttaccaacgtctacgccgattctttcgtgatcaggggcgacgaggtgcgccagatcgcccccggccagacaggcaagatcgcagactacaattataagctgccagacgatttcaccggctgcgtgatcgcctggaacagcaacaatctggattccaaagtgggcggcaactacaattatctgtaccggctgtttagaaagagcaatctgaagcccttcgagagggacatctctacagaaatctaccaggccggcagcaccccttgcaatggcgtggagggctttaactgttatttcccactccagtcctacggcttccagcccacaaacggcgtgggctatcagccttaccgcgtggtggtgctgagctttgagctgctgcacgccccagcaacagtgtgcggccccaagaagtccaccaatctggtgaagaacaagtgcgtgaacttcaacttcaacggcctgaccggcacaggcgtgctgaccgagtccaacaagaagttcctgccatttcagcagttcggcagggacatcgcagataccacagacgccgtgcgcgacccacagaccctggagatcctggacatcacaccctgctctttcggcggcgtgagcgtgatcacacccggcaccaatacaagcaaccaggtggccgtgctgtatcaggacgtgaattgtaccgaggtgcccgtggctatccacgccgatcagctgaccccaacatggcgggtgtacagcaccggctccaacgtcttccagacaagagccggatgcctgatcggagcagagcacgtgaacaattcctatgagtgcgacatcccaatcggcgccggcatctgtgcctcttaccagacccagacaaactctcccagaagagcccggagcgtggcctcccagtctatcatcgcctataccatgtccctgggcgccgag

gtgaccctggccgatgccggcttcatcaagcagtatggcgattgcctgggcgacatcgcagccagggacctgatctgcgcccagaagtttaatggcctgaccgtgctgccacccctgctgacagatgagatgatcgcacagtacacaagcgccctgctggccggcaccatcacatccggatggaccttcggcgcaggagccgccctccagatcccctttgccatgcagatggcctataggttcaacggcatcggcgtgacccagaatgtgctgtacgagaaccagaagctgatcgccaatcagtttaactccgccatcggcaagatccaggacagcctgtcctctacagccagcgccctgggcaagctccaggatgtggtgaatcagaacgcccaggccctgaataccctggtgaagcagctgagcagcaacttcggcgccatctctagcgtgctgaatgacatcctgagccggctggacaaggtggaggcagaggtgcagatcgaccggctgatcaccggccggctccagagcctccagacctatgtgacacagcagctgatcagggccgccgagatcagggccagcgccaatctggcagcaaccaagatgtccgagtgcgtgctgggccagtctaagagagtggacttttgtggcaagggctatcacctgatgtccttccctcagtctgccccacacggcgtggtgtttctgcacgtgacc

ggctgggcttcatcgccggcctgatcgccatcgtgatggtgaccatcatgctgtgctgtatgacatcctgctgttcttgcctgaagggctgctgtagctgtggctcctgctgtaagtttgacgaggatgactctgaacctgtgctgaagggcgtgaagctgcattacacctaa (SEQ ID NO: 5)
Белковая последовательность S-(deg-S2) для штамма Wuhan (от N-конца к C-концу):
MFVFLVLLPLVSSQCVNLTTRTQLPPAYTNSFTRGVYYPDKVFRSSVLHSTQDLFLPFFSNVTWFHAIHVSGTNGTKRFDNPVLPFNDGVYFASTEKSNIIRGWIFGTTLDSKTQSLLIVNNATNVVIKVCEFQFCNDPFLGVYYHKNNKSWMESEFRVYSSANNCTFEYVSQPFLMDLEGKQGNFKNLREFVFKNIDGYFKIYSKHTPINLVRDLPQGFSALEPLVDLPIGINITRFQTLLALHRSYLTPGDSSSGWTAGAAAYYVGYLQPRTFLLKYNENGTITDAVDCALDPLSETKCTLKSFTVEKGIYQTSNFRVQPTESIVRFPNITNLCPFGEVFNATRFASVYAWNRKRISNCVADYSVLYNSASFSTFKCYGVSPTKLNDLCFTNVYADSFVIRGDEVRQIAPGQTGKIADYNYKLPDDFTGCVIAWNSNNLDSKVGGNYNYLYRLFRKSNLKPFERDISTEIYQAGSTPCNGVEGFNCYFPLQSYGFQPTNGVGYQPYRVVVLSFELLHAPATVCGPKKSTNLVKNKCVNFNFNGLTGTGVLTESNKKFLPFQQFGRDIADTTDAVRDPQTLEILDITPCSFGGVSVITPGTNTSNQVAVLYQDVNCTEVPVAIHADQLTPTWRVYSTGSNVFQTRAGCLIG

QYGDCLGDIAARDLICAQKFNGLTVLPPLLTDEMIAQYTSALLAGTITSGWTFGAGAALQIPFAMQMAYRFNGIGVTQNVLYENQKLIANQFNSAIGKIQDSLSSTASALGKLQDVVNQNAQALNTLVKQLSSNFGAISSVLNDILSRLDPPEAEVQIDRLITGRLQSLQTYVTQQLIRAAEIRASANLAATKMSECVLGQSKRVDFCGKGYHLMSFPQSAPHGVVFLHVTYVPAQE

SCCKFDEDDSEPVLKGVKLHYT (SEQ ID NO: 6)
Последовательность ДНК или РНК S-(deg-S2) для штамма дельта (от 5'-конца к 3'-концу):
atgttcgtcttcctggtcctgctgcctctggtctcctcacagtgcgtcaatctgcgaactcggactcagctgccacctgcttatactaatagcttcaccagaggcgtgtactatcctgacaaggtgtttagaagctccgtgctgcactctacacaggatctgtttctgccattctttagcaacgtgacctggttccacgccatccacgtgagcggcaccaatggcacaaagcggttcgacaatcccgtgctgccttttaacgatggcgtgtacttcgcctctatcgagaagagcaacatcatcagaggctggatctttggcaccacactggactccaagacacagtctctgctgatcgtgaacaatgccaccaacgtggtcatcaaggtgtgcgagttccagttttgtaatgatcccttcctggacgtgtactatcacaagaacaataagagctggatggagtccggagtgtattctagcgccaacaactgcacatttgagtacgtgagccagcctttcctgatggacctggagggcaagcagggcaatttcaagaacctgagggagttcgtgtttaagaatatcgacggctacttcaaaatctactctaagcacacccccatcaacctggtgcgcgacctgcctcagggcttcagcgccctggagcccctggtggatctgcctatcggcatcaacatcacccggtttcagacactgctggccctgcacagaagctacctgacacccggcgactcctctagcggatggaccgccggcgctgccgcctactatgtgggctacctccagccccggaccttcctgctgaagtacaacgagaatggcaccatcacagacgcagtggattgcgccctggaccccctgagcgagacaaagtgtacactgaagtcctttaccgtggagaagggcatctatcagacatccaatttcagggtgcagccaaccgagtctatcgtgcgctttcctaatatcacaaacctgtgcccatttggcgaggtgttcaacgcaacccgcttcgccagcgtgtacgcctggaataggaagcggatcagcaactgcgtggccgactatagcgtgctgtacaactccgcctctttcagcacctttaagtgctatggcgtgtcccccacaaagctgaatgacctgtgctttaccaacgtctacgccgattctttcgtgatcaggggcgacgaggtgcgccagatcgcccccggccagacaggcaagatcgcagactacaattataagctgccagacgatttcaccggctgcgtgatcgcctggaacagcaacaatctggattccaaagtgggcggcaactacaattatcggtaccggctgtttagaaagagcaatctgaagcccttcgagagggacatctctacagaaatctaccaggccggcagcaagccttgcaatggcgtggagggctttaactgttatttcccactccagtcctacggcttccagcccacaaacggcgtgggctatcagccttaccgcgtggtggtgctgagctttgagctgctgcacgccccagcaacagtgtgcggccccaagaagtccaccaatctggtgaagaacaagtgcgtgaacttcaacttcaacggcctgaccggcacaggcgtgctgaccgagtccaacaagaagttcctgccatttcagcagttcggcagggacatcgcagataccacagacgccgtgcgcgacccacagaccctggagatcctggacatcacaccctgctctttcggcggcgtgagcgtgatcacacccggcaccaatacaagcaaccaggtggccgtgctgtatcagggcgtgaattgtaccgaggtgcccgtggctatccacgccgatcagctgaccccaacatggcgggtgtacagcaccggctccaacgtcttccagacaagagccggatgcctgatcggagcagagcacgtgaacaattcctatgagtgcgacatcccaatcggcgccggcatctgtgcctcttaccagacccagacaaactctcgcagaagagcccggagcgtggcctcccagtctatcatcgcctataccatgtccctgggcgccgagaacagcgtggcctactctcagaatagcatcgccatcccaacccagttcacaatctctgtgaccacagagatcctgcccgtgtccatgaccaagacatctgtggactgcacaatgtatatctgtggcgattctaccgagtgcagcaacctgctgctccagtacggcagcttttgtacccagctgaatagagccctgacaggcatcgccgtggagcaggataagaacacacaggaggtgttcgcccaggtgaagcaaatctacaagaccccccctatcaaggactttggcggcttccaattttcccagatcctgcctgatccatccaagccttctaagcggagctttatcgaggacctgctgttcaacaaggtgaccctggccgatgccggcttcatcaagcagtatggcgattgcctgggcgacatcgcagccagggacctgatctgcgcccagaagtttaatggcctgaccgtgctgccacccctgctgacagatgagatgatcgcacagtacacaagcgccctgctggccggcaccatcacatccggatggaccttcggcgcaggagccgccctccagatcccctttgccatgcagatggcctataggttcaacggcatcggcgtgacccagaatgtgctgtacgagaaccagaagctgatcgccaatcagtttaactccgccatcggcaagatccaggacagcctgtcctctacagccagcgccctgggcaagctccagaatgtggtgaatcagaacgcccaggccctgaataccctggtgaagcagctgagcagcaacttcggcgccatctctagcgtgctgaatgacatcctgagccggctggacaaggtggaggcagaggtgcagatcgaccggctgatcaccggccggctccagagcctccagacctatgtgacacagcagctgatcagggccgccgagatcagggccagcgccaatctggcagcaaccaagatgtccgagtgcgtgctgggccagtctaagagagtggacttttgtggcaagggctatcacctgatgtccttccctcagtctgccccacacggcgtggtgtttctgcacgtgacctacgtgcccgcccaggagaagcagttcaccacagcccctgccatctgccacgatggcaaggcccactttccaagggagggcgtgttcgtgtcccagggcacccactggtttgtgacacagcgcaatttctacgagccccagatcatcaccacagacaacaccttcgtgagcggcaactgtgacgtggtcatcggcatcgtgcagaataccgtgtatgatccactccagcccgagctggacagctttaaggaggagctggataagtatttcaagcaacacacctcccctgacgtggatctgggcgacatcagcggcatccaagcctccgtggtgaacatccagaaggagatcgaccgcctgaacgaggtggctaagaatctgcaggagagcctgatcgacctccaggagctgggcaagtatgagcagtacatcaagtggccctggtacatctggctgggcttcatcgccggcctgatcgccatcgtgatggtgaccatcatgctgtgctgtatgacatcctgctgttcttgcctgaagggctgctgtagctgtggctcctgctgtaagtttgacgaggatgactctgaacctgtgctgaagggcgtgaagctgcattacacctaa(SEQ ID NO: 27)
Белковая последовательность S-(deg-S2) для штамма дельта (от N-конца к C-концу):
MFVFLVLLPLVSSQCVNLRTRTQLPPAYTNSFTRGVYYPDKVFRSSVLHSTQDLFLPFFSNVTWFHAIHVSGTNGTKRFDNPVLPFNDGVYFASIEKSNIIRGWIFGTTLDSKTQSLLIVNNATNVVIKVCEFQFCNDPFLDVYYHKNNKSWMESGVYSSANNCTFEYVSQPFLMDLEGKQGNFKNLREFVFKNIDGYFKIYSKHTPINLVRDLPQGFSALEPLVDLPIGINITRFQTLLALHRSYLTPGDSSSGWTAGAAAYYVGYLQPRTFLLKYNENGTITDAVDCALDPLSETKCTLKSFTVEKGIYQTSNFRVQPTESIVRFPNITNLCPFGEVFNATRFASVYAWNRKRISNCVADYSVLYNSASFSTFKCYGVSPTKLNDLCFTNVYADSFVIRGDEVRQIAPGQTGKIADYNYKLPDDFTGCVIAWNSNNLDSKVGGNYNYRYRLFRKSNLKPFERDISTEIYQAGSKPCNGVEGFNCYFPLQSYGFQPTNGVGYQPYRVVVLSFELLHAPATVCGPKKSTNLVKNKCVNFNFNGLTGTGVLTESNKKFLPFQQFGRDIADTTDAVRDPQTLEILDITPCSFGGVSVITPGTNTSNQVAVLYQGVNCTEVPVAIHADQLTPTWRVYSTGSNVFQTRAGCLIGAEHVNNSYECDIPIGAGICASYQTQTNSRRRARSVASQSIIAYTMSLGAENSVAYSQNSIAIPTQFTISVTTEILPVSMTKTSVDCTMYICGDSTECSNLLLQYGSFCTQLNRALTGIAVEQDKNTQEVFAQVKQIYKTPPIKDFGGFQFSQILPDPSKPSKRSFIEDLLFNKVTLADAGFIKQYGDCLGDIAARDLICAQKFNGLTVLPPLLTDEMIAQYTSALLAGTITSGWTFGAGAALQIPFAMQMAYRFNGIGVTQNVLYENQKLIANQFNSAIGKIQDSLSSTASALGKLQNVVNQNAQALNTLVKQLSSNFGAISSVLNDILSRLDKVEAEVQIDRLITGRLQSLQTYVTQQLIRAAEIRASANLAATKMSECVLGQSKRVDFCGKGYHLMSFPQSAPHGVVFLHVTYVPAQEKQFTTAPAICHDGKAHFPREGVFVSQGTHWFVTQRNFYEPQIITTDNTFVSGNCDVVIGIVQNTVYDPLQPELDSFKEELDKYFKQHTSPDVDLGDISGIQASVVNIQKEIDRLNEVAKNLQESLIDLQELGKYEQYIKWPWYIWLGFIAGLIAIVMVTIMLCCMTSCCSCLKGCCSCGSCCKFDEDDSEPVLKGVKLHYT(SEQ ID NO: 28)
Последовательность ДНК или РНК S-(deg-S2) для Wuhan S-2P (от 5'-конца к 3'-концу):
atgttcgtcttcctggtcctgctgcctctggtctcctcacagtgcgtcaatctgacaactcggactcagctgccacctgcttatactaatagcttcaccagaggcgtgtactatcctgacaaggtgtttagaagctccgtgctgcactctacacaggatctgtttctgccattctttagcaacgtgacctggttccacgccatccacgtgagcggcaccaatggcacaaagcggttcgacaatcccgtgctgccttttaacgatggcgtgtacttcgcctctaccgagaagagcaacatcatcagaggctggatctttggcaccacactggactccaagacacagtctctgctgatcgtgaacaatgccaccaacgtggtcatcaaggtgtgcgagttccagttttgtaatgatcccttcctgggcgtgtactatcacaagaacaataagagctggatggagtccgagtttagagtgtattctagcgccaacaactgcacatttgagtacgtgagccagcctttcctgatggacctggagggcaagcagggcaatttcaagaacctgagggagttcgtgtttaagaatatcgacggctacttcaaaatctactctaagcacacccccatcaacctggtgcgcgacctgcctcagggcttcagcgccctggagcccctggtggatctgcctatcggcatcaacatcacccggtttcagacactgctggccctgcacagaagctacctgacacccggcgactcctctagcggatggaccgccggcgctgccgcctactatgtgggctacctccagccccggaccttcctgctgaagtacaacgagaatggcaccatcacagacgcagtggattgcgccctggaccccctgagcgagacaaagtgtacactgaagtcctttaccgtggagaagggcatctatcagacatccaatttcagggtgcagccaaccgagtctatcgtgcgctttcctaatatcacaaacctgtgcccatttggcgaggtgttcaacgcaacccgcttcgccagcgtgtacgcctggaataggaagcggatcagcaactgcgtggccgactatagcgtgctgtacaactccgcctctttcagcacctttaagtgctatggcgtgtcccccacaaagctgaatgacctgtgctttaccaacgtctacgccgattctttcgtgatcaggggcgacgaggtgcgccagatcgcccccggccagacaggcaagatcgcagactacaattataagctgccagacgatttcaccggctgcgtgatcgcctggaacagcaacaatctggattccaaagtgggcggcaactacaattatctgtaccggctgtttagaaagagcaatctgaagcccttcgagagggacatctctacagaaatctaccaggccggcagcaccccttgcaatggcgtggagggctttaactgttatttcccactccagtcctacggcttccagcccacaaacggcgtgggctatcagccttaccgcgtggtggtgctgagctttgagctgctgcacgccccagcaacagtgtgcggccccaagaagtccaccaatctggtgaagaacaagtgcgtgaacttcaacttcaacggcctgaccggcacaggcgtgctgaccgagtccaacaagaagttcctgccatttcagcagttcggcagggacatcgcagataccacagacgccgtgcgcgacccacagaccctggagatcctggacatcacaccctgctctttcggcggcgtgagcgtgatcacacccggcaccaatacaagcaaccaggtggccgtgctgtatcaggacgtgaattgtaccgaggtgcccgtggctatccacgccgatcagctgaccccaacatggcgggtgtacagcaccggctccaacgtcttccagacaagagccggatgcctgatcggagcagagcacgtgaacaattcctatgagtgcgacatcccaatcggcgccggcatctgtgcctcttaccagacccagacaaactctcccagaagagcccggagcgtggcctcccagtctatcatcgcctataccatgtccctgggcgccgagaacagcgtggcctactctcagaatagcatcgccatcccaacccagttcacaatctctgtgaccacagagatcctgcccgtgtccatgaccaagacatctgtggactgcacaatgtatatctgtggcgattctaccgagtgcagcaacctgctgctccagtacggcagcttttgtacccagctgaatagagccctgacaggcatcgccgtggagcaggataagaacacacaggaggtgttcgcccaggtgaagcaaatctacaagaccccccctatcaaggactttggcggcttccaattttcccagatcctgcctgatccatccaagccttctaagcggagctttatcgaggacctgctgttcaacaaggtgaccctggccgatgccggcttcatcaagcagtatggcgattgcctgggcgacatcgcagccagggacctgatctgcgcccagaagtttaatggcctgaccgtgctgccacccctgctgacagatgagatgatcgcacagtacacaagcgccctgctggccggcaccatcacatccggatggaccttcggcgcaggagccgccctccagatcccctttgccatgcagatggcctataggttcaacggcatcggcgtgacccagaatgtgctgtacgagaaccagaagctgatcgccaatcagtttaactccgccatcggcaagatccaggacagcctgtcctctacagccagcgccctgggcaagctccaggatgtggtgaatcagaacgcccaggccctgaataccctggtgaagcagctgagcagcaacttcggcgccatctctagcgt
gacctatgtgacacagcagctgatcagggccgccgagatcagggccagcgccaatctggcagcaaccaagatgtccgagtgcgtgctgggccagtctaagagagtggacttttgtggcaagggctatcacctgatgtccttccctcagtctgccccacacggcgtggtgtttctgcacgtgacctacgtgcccgcccaggagaagcagttcaccacagcccctgccatctgccacgatggcaaggcccactttccaagggagggcgtgttcgtgtcccagggcacccactggtttgtgacacagcgcaatttctacgagccccagatcatcaccacagacaacaccttcgtgagcggcaactgtgacgtggtcatcggcatcgtgcagaataccgtgtatgatccactccagcccgagctggacagctttaaggaggagctggataagtatttcaagcaacacacctcccctgacgtggatctgggcgacatcagcggcatccaagcctccgtggtgaacatccagaaggagatcgaccgcctgaacgaggtggctaagaatctgcaggagagcctgatcgacctccaggagctgggcaagtatgagcagtacatcaagtggccctggtacatctggctgggcttcatcgccggcctgatcgccatcgtgatggtgaccatcatgctgtgctgtatgacatcctgctgttcttgcctgaagggctgctgtagctgtggctcctgctgtaagtttgacgaggatgactctgaacctgtgctgaagggcgtgaagctgcattacacctaa(SEQ ID NO: 29)
Белковая последовательность S-(deg-S2) для Wuhan S-2P (от N-конца к C-концу):
MFVFLVLLPLVSSQCVNLTTRTQLPPAYTNSFTRGVYYPDKVFRSSVLHSTQDLFLPFFSNVTWFHAIHVSGTNGTKRFDNPVLPFNDGVYFASTEKSNIIRGWIFGTTLDSKTQSLLIVNNATNVVIKVCEFQFCNDPFLGVYYHKNNKSWMESEFRVYSSANNCTFEYVSQPFLMDLEGKQGNFKNLREFVFKNIDGYFKIYSKHTPINLVRDLPQGFSALEPLVDLPIGINITRFQTLLALHRSYLTPGDSSSGWTAGAAAYYVGYLQPRTFLLKYNENGTITDAVDCALDPLSETKCTLKSFTVEKGIYQTSNFRVQPTESIVRFPNITNLCPFGEVFNATRFASVYAWNRKRISNCVADYSVLYNSASFSTFKCYGVSPTKLNDLCFTNVYADSFVIRGDEVRQIAPGQTGKIADYNYKLPDDFTGCVIAWNSNNLDSKVGGNYNYLYRLFRKSNLKPFERDISTEIYQAGSTPCNGVEGFNCYFPLQSYGFQPTNGVGYQPYRVVVLSFELLHAPATVCGPKKSTNLVKNKCVNFNFNGLTGTGVLTESNKKFLPFQQFGRDIADTTDAVRDPQTLEILDITPCSFGGVSVITPGTNTSNQVAVLYQDVNCTEVPVAIHADQLTPTWRVYSTGSNVFQTRAGCLIGAEHVNNSYECDIPIGAGICASYQTQTNSPRRARSVASQSIIAYTMSLGAENSVAYSQNSIAIPTQFTISVTTEILPVSMTKTSVDCTMYICGDSTECSNLLLQYGSFCTQLNRALTGIAVEQDKNTQEVFAQVKQIYKTPPIKDFGGFQFSQILPDPSKPSKRSFIEDLLFNKVTLADAGFIKQYGDCLGDIAARDLICAQKFNGLTVLPPLLTDEMIAQYTSALLAGTITSGWTFGAGAALQIPFAMQMAYRFNGIGVTQNVLYENQKLIANQFNSAIGKIQDSLSSTASALGKLQDVVN
AAEIRASANLAATKMSECVLGQSKRVDFCGKGYHLMSFPQSAPHGVVFLHVTYVPAQEKQFTTAPAICHDGKAHFPREGVFVSQGTHWFVTQRNFYEPQIITTDNTFVSGNCDVVIGIVQNTVYDPLQPELDSFKEELDKYFKQHTSPDVDLGDISGIQASVVNIQKEIDRLNEVAKNLQESLIDLQELGKYEQYIKWPWYIWLGFIAGLIAIVMVTIMLCCMTSCCSCLKGCCSCGSCCKFDEDDSEPVLKGVKLHYT(SEQ ID NO: 30)
Последовательность ДНК или РНК S-(deg-S2) для штамма дельта S-2P (от 5'-конца к 3'-концу):
atgttcgtcttcctggtcctgctgcctctggtctcctcacagtgcgtcaatctgcgaactcggactcagctgccacctgcttatactaatagcttcaccagaggcgtgtactatcctgacaaggtgtttagaagctccgtgctgcactctacacaggatctgtttctgccattctttagcaacgtgacctggttccacgccatccacgtgagcggcaccaatggcacaaagcggttcgacaatcccgtgctgccttttaacgatggcgtgtacttcgcctctatcgagaagagcaacatcatcagaggctggatctttggcaccacactggactccaagacacagtctctgctgatcgtgaacaatgccaccaacgtggtcatcaaggtgtgcgagttccagttttgtaatgatcccttcctggacgtgtactatcacaagaacaataagagctggatggagtccggagtgtattctagcgccaacaactgcacatttgagtacgtgagccagcctttcctgatggacctggagggcaagcagggcaatttcaagaacctgagggagttcgtgtttaagaatatcgacggctacttcaaaatctactctaagcacacccccatcaacctggtgcgcgacctgcctcagggcttcagcgccctggagcccctggtggatctgcctatcggcatcaacatcacccggtttcagacactgctggccctgcacagaagctacctgacacccggcgactcctctagcggatggaccgccggcgctgccgcctactatgtgggctacctccagccccggaccttcctgctgaagtacaacgagaatggcaccatcacagacgcagtggattgcgccctggaccccctgagcgagacaaagtgtacactgaagtcctttaccgtggagaagggcatctatcagacatccaatttcagggtgcagccaaccgagtctatcgtgcgctttcctaatatcacaaacctgtgcccatttggcgaggtgttcaacgcaacccgcttcgccagcgtgtacgcctggaataggaagcggatcagcaactgcgtggccgactatagcgtgctgtacaactccgcctctttcagcacctttaagtgctatggcgtgtcccccacaaagctgaatgacctgtgctttaccaacgtctacgccgattctttcgtgatcaggggcgacgaggtgcgccagatcgcccccggccagacaggcaagatcgcagactacaattataagctgccagacgatttcaccggctgcgtgatcgcctggaacagcaacaatctggattccaaagtgggcggcaactacaattatcggtaccggctgtttagaaagagcaatctgaagcccttcgagagggacatctctacagaaatctaccaggccggcagcaagccttgcaatggcgtggagggctttaactgttatttcccactccagtcctacggcttccagcccacaaacggcgtgggctatcagccttaccgcgtggtggtgctgagctttgagctgctgcacgccccagcaacagtgtgcggccccaagaagtccaccaatctggtgaagaacaagtgcgtgaacttcaacttcaacggcctgaccggcacaggcgtgctgaccgagtccaacaagaagttcctgccatttcagcagttcggcagggacatcgcagataccacagacgccgtgcgcgacccacagaccctggagatcctggacatcacaccctgctctttcggcggcgtgagcgtgatcacacccggcaccaatacaagcaaccaggtggccgtgctgtatcagggcgtgaattgtaccgaggtgcccgtggctatccacgccgatcagctgaccccaacatggcgggtgtacagcaccggctccaacgtcttccagacaagagccggatgcctgatcggagcagagcacgtgaacaattcctatgagtgcgacatcccaatcggcgccggcatctgtgcctcttaccagacccagacaaactctcgcagaagagcccggagcgtggcctcccagtctatcatcgcctataccatgtccctgggcgccgagaacagcgtggcctactctcagaatagcatcgccatcccaacccagttcacaatctctgtgaccacagagatcctgcccgtgtccatgaccaagacatctgtggactgcacaatgtatatctgtggcgattctaccgagtgcagcaacctgctgctccagtacggcagcttttgtacccagctgaatagagccctgacaggcatcgccgtggagcaggataagaacacacaggaggtgttcgcccaggtgaagcaaatctacaagaccccccctatcaaggactttggcggcttccaattttcccagatcctgcctgatccatccaagccttctaagcggagctttatcgaggacctgctgttcaacaaggtgaccctggccgatgccggcttcatcaagcagtatggcgattgcctgggcgacatcgcagccagggacctgatctgcgcccagaagtttaatggcctgaccgtgctgccacccctgctgacagatgagatgatcgcacagtacacaagcgccctgctggccggcaccatcacatccggatggaccttcggcgcaggagccgccctccagatcccctttgccatgcagatggcctataggttcaacggcatcggcgtgacccagaatgtgctgtacgagaaccagaagctgatcgccaatcagtttaactccgccatcggcaagatccaggacagcctgtcctctacagccagcgccctgggcaagctccagaatgtggtgaatcagaacgcccaggccctgaataccctggtgaagcagctgagcagcaacttcggcgccatctctagcgtg
acctatgtgacacagcagctgatcagggccgccgagatcagggccagcgccaatctggcagcaaccaagatgtccgagtgcgtgctgggccagtctaagagagtggacttttgtggcaagggctatcacctgatgtccttccctcagtctgccccacacggcgtggtgtttctgcacgtgacctacgtgcccgcccaggagaagcagttcaccacagcccctgccatctgccacgatggcaaggcccactttccaagggagggcgtgttcgtgtcccagggcacccactggtttgtgacacagcgcaatttctacgagccccagatcatcaccacagacaacaccttcgtgagcggcaactgtgacgtggtcatcggcatcgtgcagaataccgtgtatgatccactccagcccgagctggacagctttaaggaggagctggataagtatttcaagcaacacacctcccctgacgtggatctgggcgacatcagcggcatccaagcctccgtggtgaacatccagaaggagatcgaccgcctgaacgaggtggctaagaatctgcaggagagcctgatcgacctccaggagctgggcaagtatgagcagtacatcaagtggccctggtacatctggctgggcttcatcgccggcctgatcgccatcgtgatggtgaccatcatgctgtgctgtatgacatcctgctgttcttgcctgaagggctgctgtagctgtggctcctgctgtaagtttgacgaggatgactctgaacctgtgctgaagggcgtgaagctgcattacacctaa(SEQ ID NO: 31)
Белковая последовательность S-(deg-S2) для штамма дельта S-2P (от N-конца к C-концу):
MFVFLVLLPLVSSQCVNLRTRTQLPPAYTNSFTRGVYYPDKVFRSSVLHSTQDLFLPFFSNVTWFHAIHVSGTNGTKRFDNPVLPFNDGVYFASIEKSNIIRGWIFGTTLDSKTQSLLIVNNATNVVIKVCEFQFCNDPFLDVYYHKNNKSWMESGVYSSANNCTFEYVSQPFLMDLEGKQGNFKNLREFVFKNIDGYFKIYSKHTPINLVRDLPQGFSALEPLVDLPIGINITRFQTLLALHRSYLTPGDSSSGWTAGAAAYYVGYLQPRTFLLKYNENGTITDAVDCALDPLSETKCTLKSFTVEKGIYQTSNFRVQPTESIVRFPNITNLCPFGEVFNATRFASVYAWNRKRISNCVADYSVLYNSASFSTFKCYGVSPTKLNDLCFTNVYADSFVIRGDEVRQIAPGQTGKIADYNYKLPDDFTGCVIAWNSNNLDSKVGGNYNYRYRLFRKSNLKPFERDISTEIYQAGSKPCNGVEGFNCYFPLQSYGFQPTNGVGYQPYRVVVLSFELLHAPATVCGPKKSTNLVKNKCVNFNFNGLTGTGVLTESNKKFLPFQQFGRDIADTTDAVRDPQTLEILDITPCSFGGVSVITPGTNTSNQVAVLYQGVNCTEVPVAIHADQLTPTWRVYSTGSNVFQTRAGCLIGAEHVNNSYECDIPIGAGICASYQTQTNSRRRARSVASQSIIAYTMSLGAENSVAYSQNSIAIPTQFTISVTTEILPVSMTKTSVDCTMYICGDSTECSNLLLQYGSFCTQLNRALTGIAVEQDKNTQEVFAQVKQIYKTPPIKDFGGFQFSQILPDPSKPSKRSFIEDLLFNKVTLADAGFIKQYGDCLGDIAARDLICAQKFNGLTVLPPLLTDEMIAQYTSALLAGTITSGWTFGAGAALQIPFAMQMAYRFNGIGVTQNVLYENQKLIANQFNSAIGKIQDSLSSTASALGKLQNVVNQN

EIRASANLAATKMSECVLGQSKRVDFCGKGYHLMSFPQSAPHGVVFLHVTYVPAQEKQFTTAPAICHDGKAHFPREGVFVSQGTHWFVTQRNFYEPQIITTDNTFVSGNCDVVIGIVQNTVYDPLQPELDSFKEELDKYFKQHTSPDVDLGDISGIQASVVNIQKEIDRLNEVAKNLQESLIDLQELGKYEQYIKWPWYIWLGFIAGLIAIVMVTIMLCCMTSCCSCLKGCCSCGSCCKFDEDDSEPVLKGVKLHYT(SEQ ID NO: 32)
Последовательность ДНК или РНК S-(S2-1194) для штамма Wuhan (от 5'-конца к 3'-концу):
atgttcgtcttcctggtcctgctgcctctggtctcctcacagtgcgtcaatctgacaactcggactcagctgccacctgcttatactaatagcttcaccagaggcgtgtactatcctgacaaggtgtttagaagctccgtgctgcactctacacaggatctgtttctgccattctttagcaacgtgacctggttccacgccatccacgtgagcggcaccaatggcacaaagcggttcgacaatcccgtgctgccttttaacgatggcgtgtacttcgcctctaccgagaagagcaacatcatcagaggctggatctttggcaccacactggactccaagacacagtctctgctgatcgtgaacaatgccaccaacgtggtcatcaaggtgtgcgagttccagttttgtaatgatcccttcctgggcgtgtactatcacaagaacaataagagctggatggagtccgagtttagagtgtattctagcgccaacaactgcacatttgagtacgtgagccagcctttcctgatggacctggagggcaagcagggcaatttcaagaacctgagggagttcgtgtttaagaatatcgacggctacttcaaaatctactctaagcacacccccatcaacctggtgcgcgacctgcctcagggcttcagcgccctggagcccctggtggatctgcctatcggcatcaacatcacccggtttcagacactgctggccctgcacagaagctacctgacacccggcgactcctctagcggatggaccgccggcgctgccgcctactatgtgggctacctccagccccggaccttcctgctgaagtacaacgagaatggcaccatcacagacgcagtggattgcgccctggaccccctgagcgagacaaagtgtacactgaagtcctttaccgtggagaagggcatctatcagacatccaatttcagggtgcagccaaccgagtctatcgtgcgctttcctaatatcacaaacctgtgcccatttggcgaggtgttcaacgcaacccgcttcgccagcgtgtacgcctggaataggaagcggatcagcaactgcgtggccgactatagcgtgctgtacaactccgcctctttcagcacctttaagtgctatggcgtgtcccccacaaagctgaatgacctgtgctttaccaacgtctacgccgattctttcgtgatcaggggcgacgaggtgcgccagatcgcccccggccagacaggcaagatcgcagactacaattataagctgccagacgatttcaccggctgcgtgatcgcctggaacagcaacaatctggattccaaagtgggcggcaactacaattatctgtaccggctgtttagaaagagcaatctgaagcccttcgagagggacatctctacagaaatctaccaggccggcagcaccccttgcaatggcgtggagggctttaactgttatttcccactccagtcctacggcttccagcccacaaacggcgtgggctatcagccttaccgcgtggtggtgctgagctttgagctgctgcacgccccagcaacagtgtgcggccccaagaagtccaccaatctggtgaagaacaagtgcgtgaacttcaacttcaacggcctgaccggcacaggcgtgctgaccgagtccaacaagaagttcctgccatttcagcagttcggcagggacatcgcagataccacagacgccgtgcgcgacccacagaccctggagatcctggacatcacaccctgctctttcggcggcgtgagcgtgatcacacccggcaccaatacaagcaaccaggtggccgtgctgtatcaggacgtgaattgtaccgaggtgcccgtggctatccacgccgatcagctgaccccaacatggcgggtgtacagcaccggctccaacgtcttccagacaagagccggatgcctgatcggagcagagcacgtgaacaattcctatgagtgcgacatcccaatcggcgccggcatctgtgcctcttaccagacccagacaaactctcccagaagagcccggagcgtggcctcccagtctatcatcgcctataccatgtccctgggcgccgag
Gacatctgtggactgcacaatgtatatctgtggcgattctaccgagtgcagcaacctgctgctccagtacggcagcttttgtacccagctgaatagagccctgacaggcatcgccgtggagcaggataagaacacacaggaggtgttcgcccaggtgaagcaaatctacaagaccccccctat
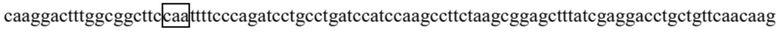
Gtgaccctggccgatgccggcttcatcaagcagtatggcgattgcctgggcgacatcgcagccagggacctgatctgcgcccagaagtttaatggcctgaccgtgctgccacccctgctgacagatgagatgatcgcacagtacacaagcgccctgctggccggcaccatcacatccggatggaccttcggcgcaggagccgccctccagatcccctttgccatgcagatggcctataggttcaacggcatcggcgtgacccagaatgtgctgtacgagaaccagaagctgatcgccaatcagtttaactccgccatcggcaagatccaggacagcctgtcctctacagccagcgccctgggcaagctccaggatgtggtgaatcagaacgcccaggccctgaataccctggtgaagcagctgagcagcaacttcggcgccatctctagcgtgctgaatgacatcctgagccggctggacaaggtggaggcagaggtgcagatcgaccggctgatcaccggccggctccagagcctccagacctatgtgacacagcagctgatcagggccgccgagatcagggccagcgccaatctggcagcaaccaagatgtccgagtgcgtgctgggccagtctaagagagtggacttttgtggcaagggctatcacctgatgtccttccctcagtctgccccacacggcgtggtgtttctgcacgtgacc

acgaggtggctaagaatctgaacgagagcctgatcgacctccaggagctgggcaagtatgagcagtacatcaagtggccctggtacatctggctgggcttcatcgccggcctgatcgccatcgtgatggtgaccatcatgctgtgctgtatgacatcctgctgttcttgcctgaagggctgctgtagctgtggctcctgctgtaagtttgacgaggatgactctgaacctgtgctgaagggcgtgaagctgcattacacctaa (SEQ ID NO: 7)
Белковая последовательность S-(S2-1194) для штамма Wuhan (от N-конца к C-концу):
MFVFLVLLPLVSSQCVNLTTRTQLPPAYTNSFTRGVYYPDKVFRSSVLHSTQDLFLPFFSNVTWFHAIHVSGTNGTKRFDNPVLPFNDGVYFASTEKSNIIRGWIFGTTLDSKTQSLLIVNNATNVVIKVCEFQFCNDPFLGVYYHKNNKSWMESEFRVYSSANNCTFEYVSQPFLMDLEGKQGNFKNLREFVFKNIDGYFKIYSKHTPINLVRDLPQGFSALEPLVDLPIGINITRFQTLLALHRSYLTPGDSSSGWTAGAAAYYVGYLQPRTFLLKYNENGTITDAVDCALDPLSETKCTLKSFTVEKGIYQTSNFRVQPTESIVRFPNITNLCPFGEVFNATRFASVYAWNRKRISNCVADYSVLYNSASFSTFKCYGVSPTKLNDLCFTNVYADSFVIRGDEVRQIAPGQTGKIADYNYKLPDDFTGCVIAWNSNNLDSKVGGNYNYLYRLFRKSNLKPFERDISTEIYQAGSTPCNGVEGFNCYFPLQSYGFQPTNGVGYQPYRVVVLSFELLHAPATVCGPKKSTNLVKNKCVNFNFNGLTGTGVLTESNKKFLPFQQFGRDIADTTDAVRDPQTLEILDITPCSFGGVSVITPGTNTSNQVAVLYQDVNCTEVPVAIHADQLTPTWRVYSTGSNVFQTRAGCLIG

QYGDCLGDIAARDLICAQKFNGLTVLPPLLTDEMIAQYTSALLAGTITSGWTFGAGAALQIPFAMQMAYRFNGIGVTQNVLYENQKLIANQFNSAIGKIQDSLSSTASALGKLQDVVNQNAQALNTLVKQLSSNFGAISSVLNDILSRLDPPEAEVQIDRLITGRLQSLQTYVTQQLIRAAEIRASANLAATKMSECVLGQSKRVDFCGKGYHLMSFPQSAPHGVVFLHVTYVPAQE

NLNESLIDLQELGKYEQYIKWPWYIWLGFIAGLIAIVMVTIMLCCMTSCCSCLKGCCCGSCCKFDEDDSEPVLKGVKLHYT (SEQ ID NO: 8)
Последовательность ДНК или РНК S-(S2-1194) штамма Wuhan S-2P (от 5'-конца к 3'-концу):
atgttcgtcttcctggtcctgctgcctctggtctcctcacagtgcgtcaatctgacaactcggactcagctgccacctgcttatactaatagcttcaccagaggcgtgtactatcctgacaaggtgtttagaagctccgtgctgcactctacacaggatctgtttctgccattctttagcaacgtgacctggttccacgccatccacgtgagcggcaccaatggcacaaagcggttcgacaatcccgtgctgccttttaacgatggcgtgtacttcgcctctaccgagaagagcaacatcatcagaggctggatctttggcaccacactggactccaagacacagtctctgctgatcgtgaacaatgccaccaacgtggtcatcaaggtgtgcgagttccagttttgtaatgatcccttcctgggcgtgtactatcacaagaacaataagagctggatggagtccgagtttagagtgtattctagcgccaacaactgcacatttgagtacgtgagccagcctttcctgatggacctggagggcaagcagggcaatttcaagaacctgagggagttcgtgtttaagaatatcgacggctacttcaaaatctactctaagcacacccccatcaacctggtgcgcgacctgcctcagggcttcagcgccctggagcccctggtggatctgcctatcggcatcaacatcacccggtttcagacactgctggccctgcacagaagctacctgacacccggcgactcctctagcggatggaccgccggcgctgccgcctactatgtgggctacctccagccccggaccttcctgctgaagtacaacgagaatggcaccatcacagacgcagtggattgcgccctggaccccctgagcgagacaaagtgtacactgaagtcctttaccgtggagaagggcatctatcagacatccaatttcagggtgcagccaaccgagtctatcgtgcgctttcctaatatcacaaacctgtgcccatttggcgaggtgttcaacgcaacccgcttcgccagcgtgtacgcctggaataggaagcggatcagcaactgcgtggccgactatagcgtgctgtacaactccgcctctttcagcacctttaagtgctatggcgtgtcccccacaaagctgaatgacctgtgctttaccaacgtctacgccgattctttcgtgatcaggggcgacgaggtgcgccagatcgcccccggccagacaggcaagatcgcagactacaattataagctgccagacgatttcaccggctgcgtgatcgcctggaacagcaacaatctggattccaaagtgggcggcaactacaattatctgtaccggctgtttagaaagagcaatctgaagcccttcgagagggacatctctacagaaatctaccaggccggcagcaccccttgcaatggcgtggagggctttaactgttatttcccactccagtcctacggcttccagcccacaaacggcgtgggctatcagccttaccgcgtggtggtgctgagctttgagctgctgcacgccccagcaacagtgtgcggccccaagaagtccaccaatctggtgaagaacaagtgcgtgaacttcaacttcaacggcctgaccggcacaggcgtgctgaccgagtccaacaagaagttcctgccatttcagcagttcggcagggacatcgcagataccacagacgccgtgcgcgacccacagaccctggagatcctggacatcacaccctgctctttcggcggcgtgagcgtgatcacacccggcaccaatacaagcaaccaggtggccgtgctgtatcaggacgtgaattgtaccgaggtgcccgtggctatccacgccgatcagctgaccccaacatggcgggtgtacagcaccggctccaacgtcttccagacaagagccggatgcctgatcggagcagagcacgtgaacaattcctatgagtgcgacatcccaatcggcgccggcatctgtgcctcttaccagacccagacaaactctcccagaagagcccggagcgtggcctcccagtctatcatcgcctataccatgtccctgggcgccgagaacagcgtggcctactctcagaatagcatcgccatcccaacccagttcacaatctctgtgaccacagagatcctgcccgtgtccatgaccaagacatctgtggactgcacaatgtatatctgtggcgattctaccgagtgcagcaacctgctgctccagtacggcagcttttgtacccagctgaatagagccctgacaggcatcgccgtggagcaggataagaacacacaggaggtgttcgcccaggtgaagcaaatctacaagaccccccctatcaaggactttggcggcttccaattttcccagatcctgcctgatccatccaagccttctaagcggagctttatcgaggacctgctgttcaacaaggtgaccctggccgatgccggcttcatcaagcagtatggcgattgcctgggcgacatcgcagccagggacctgatctgcgcccagaagtttaatggcctgaccgtgctgccacccctgctgacagatgagatgatcgcacagtacacaagcgccctgctggccggcaccatcacatccggatggaccttcggcgcaggagccgccctccagatcccctttgccatgcagatggcctataggttcaacggcatcggcgtgacccagaatgtgctgtacgagaaccagaagctgatcgccaatcagtttaactccgccatcggcaagatccaggacagcctgtcctctacagccagcgccctgggcaagctccaggatgtggtgaatcagaacgcccaggccctgaataccctggtgaagcagctgagcagcaacttcggcgccatctctagcgt
gacctatgtgacacagcagctgatcagggccgccgagatcagggccagcgccaatctggcagcaaccaagatgtccgagtgcgtgctgggccagtctaagagagtggacttttgtggcaagggctatcacctgatgtccttccctcagtctgccccacacggcgtggtgtttctgcacgtgacctacgtgcccgcccaggagaagcagttcaccacagcccctgccatctgccacgatggcaaggcccactttccaagggagggcgtgttcgtgtcccagggcacccactggtttgtgacacagcgcaatttctacgagccccagatcatcaccacagacaacaccttcgtgagcggcaactgtgacgtggtcatcggcatcgtgcagaataccgtgtatgatccactccagcccgagctggacagctttaaggaggagctggataagtatttcaagcaacacacctcccctgacgtggatctgggcgacatcagcggcatccaagcctccgtggtgaacatccagaaggagatcgaccgcctgaacgaggtggctaagaatctgaacgagagcctgatcgacctccaggagctgggcaagtatgagcagtacatcaagtggccctggtacatctggctgggcttcatcgccggcctgatcgccatcgtgatggtgaccatcatgctgtgctgtatgacatcctgctgttcttgcctgaagggctgctgtagctgtggctcctgctgtaagtttgacgaggatgactctgaacctgtgctgaagggcgtgaagctgcattacacctaa(SEQ ID NO: 33)
Белковая последовательность S-(S2-1194) для штамма Wuhan S-2P (от N-конца к C-концу):
MFVFLVLLPLVSSQCVNLTTRTQLPPAYTNSFTRGVYYPDKVFRSSVLHSTQDLFLPFFSNVTWFHAIHVSGTNGTKRFDNPVLPFNDGVYFASTEKSNIIRGWIFGTTLDSKTQSLLIVNNATNVVIKVCEFQFCNDPFLGVYYHKNNKSWMESEFRVYSSANNCTFEYVSQPFLMDLEGKQGNFKNLREFVFKNIDGYFKIYSKHTPINLVRDLPQGFSALEPLVDLPIGINITRFQTLLALHRSYLTPGDSSSGWTAGAAAYYVGYLQPRTFLLKYNENGTITDAVDCALDPLSETKCTLKSFTVEKGIYQTSNFRVQPTESIVRFPNITNLCPFGEVFNATRFASVYAWNRKRISNCVADYSVLYNSASFSTFKCYGVSPTKLNDLCFTNVYADSFVIRGDEVRQIAPGQTGKIADYNYKLPDDFTGCVIAWNSNNLDSKVGGNYNYLYRLFRKSNLKPFERDISTEIYQAGSTPCNGVEGFNCYFPLQSYGFQPTNGVGYQPYRVVVLSFELLHAPATVCGPKKSTNLVKNKCVNFNFNGLTGTGVLTESNKKFLPFQQFGRDIADTTDAVRDPQTLEILDITPCSFGGVSVITPGTNTSNQVAVLYQDVNCTEVPVAIHADQLTPTWRVYSTGSNVFQTRAGCLIGAEHVNNSYECDIPIGAGICASYQTQTNSPRRARSVASQSIIAYTMSLGAENSVAYSQNSIAIPTQFTISVTTEILPVSMTKTSVDCTMYICGDSTECSNLLLQYGSFCTQLNRALTGIAVEQDKNTQEVFAQVKQIYKTPPIKDFGGFQFSQILPDPSKPSKRSFIEDLLFNKVTLADAGFIKQYGDCLGDIAARDLICAQKFNGLTVLPPLLTDEMIAQYTSALLAGTITSGWTFGAGAALQIPFAMQMAYRFNGIGVTQNVLYENQKLIANQFNSAIGKIQDSLSSTASALGKLQDVVN
AAEIRASANLAATKMSECVLGQSKRVDFCGKGYHLMSFPQSAPHGVVFLHVTYVPAQEKQFTTAPAICHDGKAHFPREGVFVSQGTHWFVTQRNFYEPQIITTDNTFVSGNCDVVIGIVQNTVYDPLQPELDSFKEELDKYFKQHTSPDVDLGDISGIQASVVNIQKEIDRLNEVAKNLNESLIDLQELGKYEQYIKWPWYIWLGFIAGLIAIVMVTIMLCCMTSCCSCLKGCCSCGSCCKFDEDDSEPVLKGVKLHYT(SEQ ID NO: 34)
Последовательность ДНК или РНК S-(deg-RBD-801) для штамма Wuhan (от 5'-конца к 3'-концу):
Atgttcgtcttcctggtcctgctgcctctggtctcctcacagtgcgtcaatctgacaactcggactcagctgccacctgcttatactaatagcttcaccagaggcgtgtactatcctgacaaggtgtttagaagctccgtgctgcactctacacaggatctgtttctgccattctttagcaacgtgacctggttccacgccatccacgtgagcggcaccaatggcacaaagcggttcgacaatcccgtgctgccttttaacgatggcgtgtacttcgcctctaccgagaagagcaacatcatcagaggctggatctttggcaccacactggactccaagacacagtctctgctgatcgtgaacaatgccaccaacgtggtcatcaaggtgtgcgagttccagttttgtaatgatcccttcctgggcgtgtactatcacaagaacaataagagctggatggagtccgagtttagagtgtattctagcgccaacaactgcacatttgagtacgtgagccagcctttcctgatggacctggagggcaagcagggcaatttcaagaacctgagggagttcgtgtttaagaatatcgacggctacttcaaaatctactctaagcacacccccatcaacctggtgcgcgacctgcctcagggcttcagcgccctggagcccctggtggatctgcctatcggcatcaacatcacccggtttcagacactgctggccctgcacagaagctacctgacacccggcgactcctctagcggatggaccgccggcgctgccgcctactatgtgggctacctccagccccggaccttcctgctgaagtacaacgagaatggcaccatcacagacgcagtggattgcgccctggaccccctgagcgagacaaagtgtacactgaagtcctttac
gctgtacaactccgcctctttcagcacctttaagtgctatggcgtgtcccccacaaagctgaatgacctgtgctttaccaacgtctacgccgattctttcgtgatcaggggcgacgaggtgcgccagatcgcccccggccagacaggcaagatcgcagactacaattataagctgccagacgatttcaccggctgcgtgatcgcctggaacagcaacaatctggattccaaagtgggcggcaactacaattatctgtaccggctgtttagaaagagcaatctgaagcccttcgagagggacatctctacagaaatctaccaggccggcagcaccccttgcaatggcgtggagggctttaactgttatttcccactccagtcctacggcttccagcccacaaacggcgtgggctatcagccttaccgcgtggtggtgctgagctttgagctgctgcacgccccagcaacagtgtgcggccccaagaagtccaccaatctggtgaagaacaagtgcgtgaacttcaacttcaacggcctgaccggcacaggcgtgctgaccgagtccaacaagaagttcctgccatttcagcagttcggcagggacatcgcagataccacagacgccgtgcgcgacccacagaccctggagatcctggacatcacaccctgctctttcggcggcgtgagcgtgatcacacccggcaccaatacaagcaaccaggtggccgtgctgtatcaggacgtgaattgtaccgaggtgcccgtggctatccacgccgatcagctgaccccaacatggcgggtgtacagcaccggctccaacgtcttccagacaagagccggatgcctgatcggagcagagcacgtgaacaattcctatgagtgcgacatcccaatcggcgccggcatctgtgcctcttaccagacccagacaaactctcccagaagagcccggagcgtggcctcccagtctatcatcgcctataccatgtccctgggcgccgagaacagcgtggcctactctaacaatagcatcgccatcccaaccaacttcacaatctctgtgaccacagagatcctgcccgtgtccatgaccaagacatctgtggactgcacaatgtatatctgtggcgattctaccgagtgcagcaacctgctgctccagtacggcagcttttgtacccagctgaatagagccctgacaggcatcgccgtggagcaggataagaacacacaggaggtgttcgcccaggtgaagcaaatctacaagaccccc

caaggtgaccctggccgatgccggcttcatcaagcagtatggcgattgcctgggcgacatcgcagccagggacctgatctgcgcccagaagtttaatggcctgaccgtgctgccacccctgctgacagatgagatgatcgcacagtacacaagcgccctgctggccggcaccatcacatccggatggaccttcggcgcaggagccgccctccagatcccctttgccatgcagatggcctataggttcaacggcatcggcgtgacccagaatgtgctgtacgagaaccagaagctgatcgccaatcagtttaactccgccatcggcaagatccaggacagcctgtcctctacagccagcgccctgggcaagctccaggatgtggtgaatcagaacgcccaggccctgaataccctggtgaagcagctgagcagcaacttcggcgccatctctagcgtgctgaatgacatcctgagccggctggacaaggtggaggcagaggtgcagatcgaccggctgatcaccggccggctccagagcctccagacctatgtgacacagcagctgatcagggccgccgagatcagggccagcgccaatctggcagcaaccaagatgtccgagtgcgtgctgggccagtctaagagagtggacttttgtggcaagggctatcacctgatgtccttccctcagtctgccccacacggcgtggtgtttctgcacgtgacctacgtgcccgcccaggagaagaacttcaccacagcccctgccatctgccacgatggcaaggcccactttccaagggagggcgtgttcgtgtccaacggcacccactggtttgtgacacagcgcaatttctacgagccccagatcatcaccacagacaacaccttcgtgagcggcaactgtgacgtggtcatcggcatcgtgaacaataccgtgtatgatccactccagcccgagctggacagctttaaggaggagctggataagtatttcaagaatcacacctcccctgacgtggatctgggcgacatcagcggcatcaatgcctccgtggtgaacatccagaaggagatcgaccgcctgaacgaggtggctaagaatctgaacgagagcctgatcgacctccaggagctgggcaagtatgagcagtacatcaagtggccctggtacatctggctgggcttcatcgccggcctgatcgccatcgtgatggtgaccatcatgctgtgctgtatgacatcctgctgttcttgcctgaagggctgctgtagctgtggctcctgctgtaagtttgacgaggatgactctgaacctgtgctgaagggcgtgaagctgcattacacctaa (SEQ ID NO: 9)
Белковая последовательность S-(deg-RBD-801) для штамма Wuhan (от N-конца к C-концу):
MFVFLVLLPLVSSQCVNLTTRTQLPPAYTNSFTRGVYYPDKVFRSSVLHSTQDLFLPFFSNVTWFHAIHVSGTNGTKRFDNPVLPFNDGVYFASTEKSNIIRGWIFGTTLDSKTQSLLIVNNATNVVIKVCEFQFCNDPFLGVYYHKNNKSWMESEFRVYSSANNCTFEYVSQPFLMDLEGKQGNFKNLREFVFKNIDGYFKIYSKHTPINLVRDLPQGFSALEPLVDLPIGINITRFQTLLALHRSYLTPGDSSSGWTAGAAAYYVGYLQPRTFLLKYNENGTITDAVDCALD

RKRISNCVADYSVLYNSASFSTFKCYGVSPTKLNDLCFTNVYADSFVIRGDEVRQIAPGQTGKIADYNYKLPDDFTGCVIAWNSNNLDSKVGGNYNYLYRLFRKSNLKPFERDISTEIYQAGSTPCNGVEGFNCYFPLQSYGFQPTNGVGYQPYRVVVLSFELLHAPATVCGPKKSTNLVKNKCVNFNFNGLTGTGVLTESNKKFLPFQQFGRDIADTTDAVRDPQTLEILDITPCSFGGVSVITPGTNTSNQVAVLYQDVNCTEVPVAIHADQLTPTWRVYSTGSNVFQTRAGCLIGAEHVNNSYECDIPIGAGICASYQTQTNSPRRARSVASQSIIAYTMSLGAENSVAYSNNSIAIPTNFTISVTTEILPVSMTKTSVDCTMYICGDSTECSNLLLQYGSFCTQLNRALTGIAVE
KQYGDCLGDIAARDLICAQKFNGLTVLPPLLTDEMIAQYTSALLAGTITSGWTFGAGAALQIPFAMQMAYRFNGIGVTQNVLYENQKLIANQFNSAIGKIQDSLSSTASALGKLQDVVNQNAQALNTLVKQLSSNFGAISSVLNDILSRLDPPEAEVQIDRLITGRLQSLQTYVTQQLIRAAEIRASANLAATKMSECVLGQSKRVDFCGKGYHLMSFPQSAPHGVVFLHVTYVPAQEKNFTTAPAICHDGKAHFPREGVFVSNGTHWFVTQRNFYEPQIITTDNTFVSGNCDVVIGIVNNTVYDPLQPELDSFKEELDKYFKNHTSPDVDLGDISGINASVVNIQKEIDRLNEVAKNLNESLIDLQELGKYEQYIKWPWYIWLGFIAGLIAIVMVTIMLCCMTSCCSCLKGCCSCGSCCKFDEDDSEPVLKGVKLHYT (SEQ ID NO: 10)
Последовательность ДНК или РНК S-(deg-RBD-801) для штамма Wuhan S-2P (от 5'-конца к 3'-концу):
atgttcgtcttcctggtcctgctgcctctggtctcctcacagtgcgtcaatctgacaactcggactcagctgccacctgcttatactaatagcttcaccagaggcgtgtactatcctgacaaggtgtttagaagctccgtgctgcactctacacaggatctgtttctgccattctttagcaacgtgacctggttccacgccatccacgtgagcggcaccaatggcacaaagcggttcgacaatcccgtgctgccttttaacgatggcgtgtacttcgcctctaccgagaagagcaacatcatcagaggctggatctttggcaccacactggactccaagacacagtctctgctgatcgtgaacaatgccaccaacgtggtcatcaaggtgtgcgagttccagttttgtaatgatcccttcctgggcgtgtactatcacaagaacaataagagctggatggagtccgagtttagagtgtattctagcgccaacaactgcacatttgagtacgtgagccagcctttcctgatggacctggagggcaagcagggcaatttcaagaacctgagggagttcgtgtttaagaatatcgacggctacttcaaaatctactctaagcacacccccatcaacctggtgcgcgacctgcctcagggcttcagcgccctggagcccctggtggatctgcctatcggcatcaacatcacccggtttcagacactgctggccctgcacagaagctacctgacacccggcgactcctctagcggatggaccgccggcgctgccgcctactatgtgggctacctccagccccggaccttcctgctgaagtacaacgagaatggcaccatcacagacgcagtggattgcgccctggaccccctgagcgagacaaagtgtacactgaagtcctttaccgtggagaagggcatctatcagacatccaatttcagggtgcagccagccgaggcgatcgtgcgctttcctgaaatcacaaacctgtgcccatttggcgaggtgttcgaggcaacccgcttcgccagcgtgtacgcctggaataggaagcggatcagcaactgcgtggccgactatagcgtgctgtacaactccgcctctttcagcacctttaagtgctatggcgtgtcccccacaaagctgaatgacctgtgctttaccaacgtctacgccgattctttcgtgatcaggggcgacgaggtgcgccagatcgcccccggccagacaggcaagatcgcagactacaattataagctgccagacgatttcaccggctgcgtgatcgcctggaacagcaacaatctggattccaaagtgggcggcaactacaattatctgtaccggctgtttagaaagagcaatctgaagcccttcgagagggacatctctacagaaatctaccaggccggcagcaccccttgcaatggcgtggagggctttaactgttatttcccactccagtcctacggcttccagcccacaaacggcgtgggctatcagccttaccgcgtggtggtgctgagctttgagctgctgcacgccccagcaacagtgtgcggccccaagaagtccaccaatctggtgaagaacaagtgcgtgaacttcaacttcaacggcctgaccggcacaggcgtgctgaccgagtccaacaagaagttcctgccatttcagcagttcggcagggacatcgcagataccacagacgccgtgcgcgacccacagaccctggagatcctggacatcacaccctgctctttcggcggcgtgagcgtgatcacacccggcaccaatacaagcaaccaggtggccgtgctgtatcaggacgtgaattgtaccgaggtgcccgtggctatccacgccgatcagctgaccccaacatggcgggtgtacagcaccggctccaacgtcttccagacaagagccggatgcctgatcggagcagagcacgtgaacaattcctatgagtgcgacatcccaatcggcgccggcatctgtgcctcttaccagacccagacaaactctcccagaagagcccggagcgtggcctcccagtctatcatcgcctataccatgtccctgggcgccgagaacagcgtggcctactctaacaatagcatcgccatcccaaccaacttcacaatctctgtgaccacagagatcctgcccgtgtccatgaccaagacatctgtggactgcacaatgtatatctgtggcgattctaccgagtgcagcaacctgctgctccagtacggcagcttttgtacccagctgaatagagccctgacaggcatcgccgtggagcaggataagaacacacaggaggtgttcgcccaggtgaagcaaatctacaagaccccccctatcaaggactttggcggcttccaattttcccagatcctgcctgatccatccaagccttctaagcggagctttatcgaggacctgctgttcaacaaggtgaccctggccgatgccggcttcatcaagcagtatggcgattgcctgggcgacatcgcagccagggacctgatctgcgcccagaagtttaatggcctgaccgtgctgccacccctgctgacagatgagatgatcgcacagtacacaagcgccctgctggccggcaccatcacatccggatggaccttcggcgcaggagccgccctccagatcccctttgccatgcagatggcctataggttcaacggcatcggcgtgacccagaatgtgctgtacgagaaccagaagctgatcgccaatcagtttaactccgccatcggcaagatccaggacagcctgtcctctacagccagcgccctgggcaagctccaggatgtggtgaatcagaacgcccaggccctgaataccctggtgaagcagctgagcagcaacttcggcgccatctct
ctccagacctatgtgacacagcagctgatcagggccgccgagatcagggccagcgccaatctggcagcaaccaagatgtccgagtgcgtgctgggccagtctaagagagtggacttttgtggcaagggctatcacctgatgtccttccctcagtctgccccacacggcgtggtgtttctgcacgtgacctacgtgcccgcccaggagaagaacttcaccacagcccctgccatctgccacgatggcaaggcccactttccaagggagggcgtgttcgtgtccaacggcacccactggtttgtgacacagcgcaatttctacgagccccagatcatcaccacagacaacaccttcgtgagcggcaactgtgacgtggtcatcggcatcgtgaacaataccgtgtatgatccactccagcccgagctggacagctttaaggaggagctggataagtatttcaagaatcacacctcccctgacgtggatctgggcgacatcagcggcatcaatgcctccgtggtgaacatccagaaggagatcgaccgcctgaacgaggtggctaagaatctgaacgagagcctgatcgacctccaggagctgggcaagtatgagcagtacatcaagtggccctggtacatctggctgggcttcatcgccggcctgatcgccatcgtgatggtgaccatcatgctgtgctgtatgacatcctgctgttcttgcctgaagggctgctgtagctgtggctcctgctgtaagtttgacgaggatgactctgaacctgtgctgaagggcgtgaagctgcattacacctaa(SEQ ID NO: 35)
Белковая последовательность S-(deg-RBD-801) для штамма Wuhan S-2P (от N-конца к C-концу):
MFVFLVLLPLVSSQCVNLTTRTQLPPAYTNSFTRGVYYPDKVFRSSVLHSTQDLFLPFFSNVTWFHAIHVSGTNGTKRFDNPVLPFNDGVYFASTEKSNIIRGWIFGTTLDSKTQSLLIVNNATNVVIKVCEFQFCNDPFLGVYYHKNNKSWMESEFRVYSSANNCTFEYVSQPFLMDLEGKQGNFKNLREFVFKNIDGYFKIYSKHTPINLVRDLPQGFSALEPLVDLPIGINITRFQTLLALHRSYLTPGDSSSGWTAGAAAYYVGYLQPRTFLLKYNENGTITDAVDCALDPLSETKCTLKSFTVEKGIYQTSNFRVQPAEAIVRFPQITNLCPFGEVFQATRFASVYAWNRKRISNCVADYSVLYNSASFSTFKCYGVSPTKLNDLCFTNVYADSFVIRGDEVRQIAPGQTGKIADYNYKLPDDFTGCVIAWNSNNLDSKVGGNYNYLYRLFRKSNLKPFERDISTEIYQAGSTPCNGVEGFNCYFPLQSYGFQPTNGVGYQPYRVVVLSFELLHAPATVCGPKKSTNLVKNKCVNFNFNGLTGTGVLTESNKKFLPFQQFGRDIADTTDAVRDPQTLEILDITPCSFGGVSVITPGTNTSNQVAVLYQDVNCTEVPVAIHADQLTPTWRVYSTGSNVFQTRAGCLIGAEHVNNSYECDIPIGAGICASYQTQTNSPRRARSVASQSIIAYTMSLGAENSVAYSNNSIAIPTNFTISVTTEILPVSMTKTSVDCTMYICGDSTECSNLLLQYGSFCTQLNRALTGIAVEQDKNTQEVFAQVKQIYKTPPIKDFGGFQFSQILPDPSKPSKRSFIEDLLFNKVTLADAGFIKQYGDCLGDIAARDLICAQKFNGLTVLPPLLTDEMIAQYTSALLAGTITSGWTFGAGAALQIPFAMQMAYRFNGIGVTQNVLYENQKLIANQFNSAIGKIQDSLSSTASALGKLQDVV
RAAEIRASANLAATKMSECVLGQSKRVDFCGKGYHLMSFPQSAPHGVVFLHVTYVPAQEKNFTTAPAICHDGKAHFPREGVFVSNGTHWFVTQRNFYEPQIITTDNTFVSGNCDVVIGIVNNTVYDPLQPELDSFKEELDKYFKNHTSPDVDLGDISGINASVVNIQKEIDRLNEVAKNLNESLIDLQELGKYEQYIKWPWYIWLGFIAGLIAIVMVTIMLCCMTSCCSCLKGCCSCGSCCKFDEDDSEPVLKGVKLHYT(SEQ ID NO: 36)
Последовательность ДНК или РНК S-(deg-RBD-1194) штамма Wuhan (от 5'-конца к 3'-концу):
Atgttcgtcttcctggtcctgctgcctctggtctcctcacagtgcgtcaatctgacaactcggactcagctgccacctgcttatactaatagcttcaccagaggcgtgtactatcctgacaaggtgtttagaagctccgtgctgcactctacacaggatctgtttctgccattctttagcaacgtgacctggttccacgccatccacgtgagcggcaccaatggcacaaagcggttcgacaatcccgtgctgccttttaacgatggcgtgtacttcgcctctaccgagaagagcaacatcatcagaggctggatctttggcaccacactggactccaagacacagtctctgctgatcgtgaacaatgccaccaacgtggtcatcaaggtgtgcgagttccagttttgtaatgatcccttcctgggcgtgtactatcacaagaacaataagagctggatggagtccgagtttagagtgtattctagcgccaacaactgcacatttgagtacgtgagccagcctttcctgatggacctggagggcaagcagggcaatttcaagaacctgagggagttcgtgtttaagaatatcgacggctacttcaaaatctactctaagcacacccccatcaacctggtgcgcgacctgcctcagggcttcagcgccctggagcccctggtggatctgcctatcggcatcaacatcacccggtttcagacactgctggccctgcacagaagctacctgacacccggcgactcctctagcggatggaccgccggcgctgccgcctactatgtgggctacctccagccccggaccttcctgctgaagtacaacgagaatggcaccatcacagacgcagtggattgcgccctggaccccctgagcgagacaaagtgtacactgaagtcctttac
gctgtacaactccgcctctttcagcacctttaagtgctatggcgtgtcccccacaaagctgaatgacctgtgctttaccaacgtctacgccgattctttcgtgatcaggggcgacgaggtgcgccagatcgcccccggccagacaggcaagatcgcagactacaattataagctgccagacgatttcaccggctgcgtgatcgcctggaacagcaacaatctggattccaaagtgggcggcaactacaattatctgtaccggctgtttagaaagagcaatctgaagcccttcgagagggacatctctacagaaatctaccaggccggcagcaccccttgcaatggcgtggagggctttaactgttatttcccactccagtcctacggcttccagcccacaaacggcgtgggctatcagccttaccgcgtggtggtgctgagctttgagctgctgcacgccccagcaacagtgtgcggccccaagaagtccaccaatctggtgaagaacaagtgcgtgaacttcaacttcaacggcctgaccggcacaggcgtgctgaccgagtccaacaagaagttcctgccatttcagcagttcggcagggacatcgcagataccacagacgccgtgcgcgacccacagaccctggagatcctggacatcacaccctgctctttcggcggcgtgagcgtgatcacacccggcaccaatacaagcaaccaggtggccgtgctgtatcaggacgtgaattgtaccgaggtgcccgtggctatccacgccgatcagctgaccccaacatggcgggtgtacagcaccggctccaacgtcttccagacaagagccggatgcctgatcggagcagagcacgtgaacaattcctatgagtgcgacatcccaatcggcgccggcatctgtgcctcttaccagacccagacaaactctcccagaagagcccggagcgtggcctcccagtctatcatcgcctataccatgtccctgggcgccgagaacagcgtggcctactctaacaatagcatcgccatcccaaccaacttcacaatctctgtgaccacagagatcctgcccgtgtccatgaccaagacatctgtggactgcacaatgtatatctgtggcgattctaccgagtgcagcaacctgctgctccagtacggcagcttttgtacccagctgaatagagccctgacaggcatcgccgtggagcaggataagaacacacaggaggtgttcgcccaggtgaagcaaatctacaagaccccccctatcaaggactttggcggcttcaatttttcccagatcctgcctgatccatccaagccttctaagcggagctttatcgaggacctgctgttcaacaaggtgaccctggccgatgccggcttcatcaagcagtatggcgattgcctgggcgacatcgcagccagggacctgatctgcgcccagaagtttaatggcctgaccgtgctgccacccctgctgacagatgagatgatcgcacagtacacaagcgccctgctggccggcaccatcacatccggatggaccttcggcgcaggagccgccctccagatcccctttgccatgcagatggcctataggttcaacggcatcggcgtgacccagaatgtgctgtacgagaaccagaagctgatcgccaatcagtttaactccgccatcggcaagatccaggacagcctgtcctctacagccagcgccctgggcaagctccaggatgtggtgaatcagaacgcccaggccctgaataccctggtgaagcagctgagcagcaacttcggcgccatctctagcgtgctgaatgacatcctgagccggctggacaaggtggaggcagaggtgcagatcgaccggctgatcaccggccggctccagagcctccagacctatgtgacacagcagctgatcagggccgccgagatcagggccagcgccaatctggcagcaaccaagatgtccgagtgcgtgctgggccagtctaagagagtggacttttgtggcaagggctatcacctgatgtccttccctcagtctgccccacacggcgtggtgtttctgcacgtgacctacgtgcccgcccaggagaagaacttcaccacagcccctgccatctgccacgatggcaaggcccactttccaagggagggcgtgttcgtgtccaacggcacccactggtttgtgacacagcgcaatttctacgagccccagatcatcaccacagacaacaccttcgtgagcggcaactgtgacgtggtcatcggcatcgtgaacaataccgtgtatgatccactccagcccgagctggacagctttaaggaggagctggataagtatttcaagaatcacacctcccctgacgtggatctgggcgacatcagcggcatcaatgcctccgtggtgaacatccagaaggagatcgaccgc
catctggctgggcttcatcgccggcctgatcgccatcgtgatggtgaccatcatgctgtgctgtatgacatcctgctgttcttgcctgaagggctgctgtagctgtggctcctgctgtaagtttgacgaggatgactctgaacctgtgctgaagggcgtgaagctgcattacacctaa (SEQ ID NO: 11)
Белковая последовательность S-(deg-RBD-1194) для штамма Wuhan (от N-конца к C-концу):
MFVFLVLLPLVSSQCVNLTTRTQLPPAYTNSFTRGVYYPDKVFRSSVLHSTQDLFLPFFSNVTWFHAIHVSGTNGTKRFDNPVLPFNDGVYFASTEKSNIIRGWIFGTTLDSKTQSLLIVNNATNVVIKVCEFQFCNDPFLGVYYHKNNKSWMESEFRVYSSANNCTFEYVSQPFLMDLEGKQGNFKNLREFVFKNIDGYFKIYSKHTPINLVRDLPQGFSALEPLVDLPIGINITRFQTLLALHRSYLTPGDSSSGWTAGAAAYYVGYLQPRTFLLKYNENGTITDAVDCALD

RKRISNCVADYSVLYNSASFSTFKCYGVSPTKLNDLCFTNVYADSFVIRGDEVRQIAPGQTGKIADYNYKLPDDFTGCVIAWNSNNLDSKVGGNYNYLYRLFRKSNLKPFERDISTEIYQAGSTPCNGVEGFNCYFPLQSYGFQPTNGVGYQPYRVVVLSFELLHAPATVCGPKKSTNLVKNKCVNFNFNGLTGTGVLTESNKKFLPFQQFGRDIADTTDAVRDPQTLEILDITPCSFGGVSVITPGTNTSNQVAVLYQDVNCTEVPVAIHADQLTPTWRVYSTGSNVFQTRAGCLIGAEHVNNSYECDIPIGAGICASYQTQTNSPRRARSVASQSIIAYTMSLGAENSVAYSNNSIAIPTNFTISVTTEILPVSMTKTSVDCTMYICGDSTECSNLLLQYGSFCTQLNRALTGIAVEQDKNTQEVFAQVKQIYKTPPIKDFGGFNFSQILPDPSKPSKRSFIEDLLFNKVTLADAGFIKQYGDCLGDIAARDLICAQKFNGLTVLPPLLTDEMIAQYTSALLAGTITSGWTFGAGAALQIPFAMQMAYRFNGIGVTQNVLYENQKLIANQFNSAIGKIQDSLSSTASALGKLQDVVNQNAQALNTLVKQLSSNFGAISSVLNDILSRLDPPEAEVQIDRLITGRLQSLQTYVTQQLIRAAEIRASANLAATKMSECVLGQSKRVDFCGKGYHLMSFPQSAPHGVVFLHVTYVPAQEKNFTTAPAICHDGKAHFPREGVFVSNGTHWFVTQRNFYEPQIITTDNTFVSGNCDVVIGIVNNTVYDPLQPELDSFKEELDKYFKNHTSPDVDLGDISGINASVVNIQKEIDRLNEVAK
SCCKFDEDDSEPVLKGVKLHYT (SEQ ID NO: 12)
Последовательность ДНК или РНК S-(deg-RBD-1194) штамма Wuhan S-2P (от 5'-конца к 3'-концу):
atgttcgtcttcctggtcctgctgcctctggtctcctcacagtgcgtcaatctgacaactcggactcagctgccacctgcttatactaatagcttcaccagaggcgtgtactatcctgacaaggtgtttagaagctccgtgctgcactctacacaggatctgtttctgccattctttagcaacgtgacctggttccacgccatccacgtgagcggcaccaatggcacaaagcggttcgacaatcccgtgctgccttttaacgatggcgtgtacttcgcctctaccgagaagagcaacatcatcagaggctggatctttggcaccacactggactccaagacacagtctctgctgatcgtgaacaatgccaccaacgtggtcatcaaggtgtgcgagttccagttttgtaatgatcccttcctgggcgtgtactatcacaagaacaataagagctggatggagtccgagtttagagtgtattctagcgccaacaactgcacatttgagtacgtgagccagcctttcctgatggacctggagggcaagcagggcaatttcaagaacctgagggagttcgtgtttaagaatatcgacggctacttcaaaatctactctaagcacacccccatcaacctggtgcgcgacctgcctcagggcttcagcgccctggagcccctggtggatctgcctatcggcatcaacatcacccggtttcagacactgctggccctgcacagaagctacctgacacccggcgactcctctagcggatggaccgccggcgctgccgcctactatgtgggctacctccagccccggaccttcctgctgaagtacaacgagaatggcaccatcacagacgcagtggattgcgccctggaccccctgagcgagacaaagtgtacactgaagtcctttaccgtggagaagggcatctatcagacatccaatttcagggtgcagccagccgaggcgatcgtgcgctttcctgaaatcacaaacctgtgcccatttggcgaggtgttcgaggcaacccgcttcgccagcgtgtacgcctggaataggaagcggatcagcaactgcgtggccgactatagcgtgctgtacaactccgcctctttcagcacctttaagtgctatggcgtgtcccccacaaagctgaatgacctgtgctttaccaacgtctacgccgattctttcgtgatcaggggcgacgaggtgcgccagatcgcccccggccagacaggcaagatcgcagactacaattataagctgccagacgatttcaccggctgcgtgatcgcctggaacagcaacaatctggattccaaagtgggcggcaactacaattatctgtaccggctgtttagaaagagcaatctgaagcccttcgagagggacatctctacagaaatctaccaggccggcagcaccccttgcaatggcgtggagggctttaactgttatttcccactccagtcctacggcttccagcccacaaacggcgtgggctatcagccttaccgcgtggtggtgctgagctttgagctgctgcacgccccagcaacagtgtgcggccccaagaagtccaccaatctggtgaagaacaagtgcgtgaacttcaacttcaacggcctgaccggcacaggcgtgctgaccgagtccaacaagaagttcctgccatttcagcagttcggcagggacatcgcagataccacagacgccgtgcgcgacccacagaccctggagatcctggacatcacaccctgctctttcggcggcgtgagcgtgatcacacccggcaccaatacaagcaaccaggtggccgtgctgtatcaggacgtgaattgtaccgaggtgcccgtggctatccacgccgatcagctgaccccaacatggcgggtgtacagcaccggctccaacgtcttccagacaagagccggatgcctgatcggagcagagcacgtgaacaattcctatgagtgcgacatcccaatcggcgccggcatctgtgcctcttaccagacccagacaaactctcccagaagagcccggagcgtggcctcccagtctatcatcgcctataccatgtccctgggcgccgagaacagcgtggcctactctaacaatagcatcgccatcccaaccaacttcacaatctctgtgaccacagagatcctgcccgtgtccatgaccaagacatctgtggactgcacaatgtatatctgtggcgattctaccgagtgcagcaacctgctgctccagtacggcagcttttgtacccagctgaatagagccctgacaggcatcgccgtggagcaggataagaacacacaggaggtgttcgcccaggtgaagcaaatctacaagaccccccctatcaaggactttggcggcttcaatttttcccagatcctgcctgatccatccaagccttctaagcggagctttatcgaggacctgctgttcaacaaggtgaccctggccgatgccggcttcatcaagcagtatggcgattgcctgggcgacatcgcagccagggacctgatctgcgcccagaagtttaatggcctgaccgtgctgccacccctgctgacagatgagatgatcgcacagtacacaagcgccctgctggccggcaccatcacatccggatggaccttcggcgcaggagccgccctccagatcccctttgccatgcagatggcctataggttcaacggcatcggcgtgacccagaatgtgctgtacgagaaccagaagctgatcgccaatcagtttaactccgccatcggcaagatccaggacagcctgtcctctacagccagcgccctgggcaagctccaggatgtggtgaatcagaacgcccaggccctgaataccctggtgaagcagctgagcagcaacttcggcgccatctct
ctccagacctatgtgacacagcagctgatcagggccgccgagatcagggccagcgccaatctggcagcaaccaagatgtccgagtgcgtgctgggccagtctaagagagtggacttttgtggcaagggctatcacctgatgtccttccctcagtctgccccacacggcgtggtgtttctgcacgtgacctacgtgcccgcccaggagaagaacttcaccacagcccctgccatctgccacgatggcaaggcccactttccaagggagggcgtgttcgtgtccaacggcacccactggtttgtgacacagcgcaatttctacgagccccagatcatcaccacagacaacaccttcgtgagcggcaactgtgacgtggtcatcggcatcgtgaacaataccgtgtatgatccactccagcccgagctggacagctttaaggaggagctggataagtatttcaagaatcacacctcccctgacgtggatctgggcgacatcagcggcatcaatgcctccgtggtgaacatccagaaggagatcgaccgcctgaacgaggtggctaagaatctgcaggagagcctgatcgacctccaggagctgggcaagtatgagcagtacatcaagtggccctggtacatctggctgggcttcatcgccggcctgatcgccatcgtgatggtgaccatcatgctgtgctgtatgacatcctgctgttcttgcctgaagggctgctgtagctgtggctcctgctgtaagtttgacgaggatgactctgaacctgtgctgaagggcgtgaagctgcattacacctaa(SEQ ID NO: 37)
Белковая последовательность S-(deg-RBD-1194) для штамма Wuhan S-2P (от N-конца к C-концу):
MFVFLVLLPLVSSQCVNLTTRTQLPPAYTNSFTRGVYYPDKVFRSSVLHSTQDLFLPFFSNVTWFHAIHVSGTNGTKRFDNPVLPFNDGVYFASTEKSNIIRGWIFGTTLDSKTQSLLIVNNATNVVIKVCEFQFCNDPFLGVYYHKNNKSWMESEFRVYSSANNCTFEYVSQPFLMDLEGKQGNFKNLREFVFKNIDGYFKIYSKHTPINLVRDLPQGFSALEPLVDLPIGINITRFQTLLALHRSYLTPGDSSSGWTAGAAAYYVGYLQPRTFLLKYNENGTITDAVDCALDPLSETKCTLKSFTVEKGIYQTSNFRVQPAEAIVRFPQITNLCPFGEVFQATRFASVYAWNRKRISNCVADYSVLYNSASFSTFKCYGVSPTKLNDLCFTNVYADSFVIRGDEVRQIAPGQTGKIADYNYKLPDDFTGCVIAWNSNNLDSKVGGNYNYLYRLFRKSNLKPFERDISTEIYQAGSTPCNGVEGFNCYFPLQSYGFQPTNGVGYQPYRVVVLSFELLHAPATVCGPKKSTNLVKNKCVNFNFNGLTGTGVLTESNKKFLPFQQFGRDIADTTDAVRDPQTLEILDITPCSFGGVSVITPGTNTSNQVAVLYQDVNCTEVPVAIHADQLTPTWRVYSTGSNVFQTRAGCLIGAEHVNNSYECDIPIGAGICASYQTQTNSPRRARSVASQSIIAYTMSLGAENSVAYSNNSIAIPTNFTISVTTEILPVSMTKTSVDCTMYICGDSTECSNLLLQYGSFCTQLNRALTGIAVEQDKNTQEVFAQVKQIYKTPPIKDFGGFNFSQILPDPSKPSKRSFIEDLLFNKVTLADAGFIKQYGDCLGDIAARDLICAQKFNGLTVLPPLLTDEMIAQYTSALLAGTITSGWTFGAGAALQIPFAMQMAYRFNGIGVTQNVLYENQKLIANQFNSAIGKIQDSLSSTASALGKLQDVV
RAAEIRASANLAATKMSECVLGQSKRVDFCGKGYHLMSFPQSAPHGVVFLHVTYVPAQEKNFTTAPAICHDGKAHFPREGVFVSNGTHWFVTQRNFYEPQIITTDNTFVSGNCDVVIGIVNNTVYDPLQPELDSFKEELDKYFKNHTSPDVDLGDISGINASVVNIQKEIDRLNEVAKNLQESLIDLQELGKYEQYIKWPWYIWLGFIAGLIAIVMVTIMLCCMTSCCSCLKGCCSCGSCCKFDEDDSEPVLKGVKLHYT(SEQ ID NO: 38)
Последовательность ДНК или РНК S-(deg-RBD-122-165-234) штамма Wuhan (от 5'-конца к 3'-концу):
Atgttcgtcttcctggtcctgctgcctctggtctcctcacagtgcgtcaatctgacaactcggactcagctgccacctgcttatactaatagcttcaccagaggcgtgtactatcctgacaaggtgtttagaagctccgtgctgcactctacacaggatctgtttctgccattctttagcaacgtgacctggttccacgccatccacgtgagcggcaccaatggcacaaagcggttcgacaatcccgtgctgccttttaacgatggcgtgtacttcg

cgtgctgtacaactccgcctctttcagcacctttaagtgctatggcgtgtcccccacaaagctgaatgacctgtgctttaccaacgtctacgccgattctttcgtgatcaggggcgacgaggtgcgccagatcgcccccggccagacaggcaagatcgcagactacaattataagctgccagacgatttcaccggctgcgtgatcgcctggaacagcaacaatctggattccaaagtgggcggcaactacaattatctgtaccggctgtttagaaagagcaatctgaagcccttcgagagggacatctctacagaaatctaccaggccggcagcaccccttgcaatggcgtggagggctttaactgttatttcccactccagtcctacggcttccagcccacaaacggcgtgggctatcagccttaccgcgtggtggtgctgagctttgagctgctgcacgccccagcaacagtgtgcggccccaagaagtccaccaatctggtgaagaacaagtgcgtgaacttcaacttcaacggcctgaccggcacaggcgtgctgaccgagtccaacaagaagttcctgccatttcagcagttcggcagggacatcgcagataccacagacgccgtgcgcgacccacagaccctggagatcctggacatcacaccctgctctttcggcggcgtgagcgtgatcacacccggcaccaatacaagcaaccaggtggccgtgctgtatcaggacgtgaattgtaccgaggtgcccgtggctatccacgccgatcagctgaccccaacatggcgggtgtacagcaccggctccaacgtcttccagacaagagccggatgcctgatcggagcagagcacgtgaacaattcctatgagtgcgacatcccaatcggcgccggcatctgtgcctcttaccagacccagacaaactctcccagaagagcccggagcgtggcctcccagtctatcatcgcctataccatgtccctgggcgccgagaacagcgtggcctactctaacaatagcatcgccatcccaaccaacttcacaatctctgtgaccacagagatcctgcccgtgtccatgaccaagacatctgtggactgcacaatgtatatctgtggcgattctaccgagtgcagcaacctgctgctccagtacggcagcttttgtacccagctgaatagagccctgacaggcatcgccgtggagcaggataagaacacacaggaggtgttcgcccaggtgaagcaaatctacaagaccccccctatcaaggactttggcggcttcaatttttcccagatcctgcctgatccatccaagccttctaagcggagctttatcgaggacctgctgttcaacaaggtgaccctggccgatgccggcttcatcaagcagtatggcgattgcctgggcgacatcgcagccagggacctgatctgcgcccagaagtttaatggcctgaccgtgctgccacccctgctgacagatgagatgatcgcacagtacacaagcgccctgctggccggcaccatcacatccggatggaccttcggcgcaggagccgccctccagatcccctttgccatgcagatggcctataggttcaacggcatcggcgtgacccagaatgtgctgtacgagaaccagaagctgatcgccaatcagtttaactccgccatcggcaagatccaggacagcctgtcctctacagccagcgccctgggcaagctccaggatgtggtgaatcagaacgcccaggccctgaataccctggtgaagcagctgagcagcaacttcggcgccatctctagcgtgctgaatgacatcctgagccggctggacaaggtggaggcagaggtgcagatcgaccggctgatcaccggccggctccagagcctccagacctatgtgacacagcagctgatcagggccgccgagatcagggccagcgccaatctggcagcaaccaagatgtccgagtgcgtgctgggccagtctaagagagtggacttttgtggcaagggctatcacctgatgtccttccctcagtctgccccacacggcgtggtgtttctgcacgtgacctacgtgcccgcccaggagaagaacttcaccacagcccctgccatctgccacgatggcaaggcccactttccaagggagggcgtgttcgtgtccaacggcacccactggtttgtgacacagcgcaatttctacgagccccagatcatcaccacagacaacaccttcgtgagcggcaactgtgacgtggtcatcggcatcgtgaacaataccgtgtatgatccactccagcccgagctggacagctttaaggaggagctggataagtatttcaagaatcacacctcccctgacgtggatctgggcgacatcagcggcatcaatgcctccgtggtgaacatccagaaggagatcgaccgcctgaacgaggtggctaagaatctgaacgagagcctgatcgacctccaggagctgggcaagtatgagcagtacatcaagtggccctggtacatctggctgggcttcatcgccggcctgatcgccatcgtgatggtgaccatcatgctgtgctgtatgacatcctgctgttcttgcctgaagggctgctgtagctgtggctcctgctgtaagtttgacgaggatgactctgaacctgtgctgaagggcgtgaagctgcattacacctaa (SEQ ID NO: 13)
Белковая последовательность S-(deg-RBD-122-165-234) штамма Wuhan (от N-конца к C-концу):
MFVFLVLLPLVSSQCVNLTTRTQLPPAYTNSFTRGVYYPDKVFRSSVLHSTQDLFLPFFSNVTWFHAIHVSGTNGTKRFDNPVLPFNDGVYFASTEKSNIIRGWIFGTTLDSKTQS
NRKRISNCVADYSVLYNSASFSTFKCYGVSPTKLNDLCFTNVYADSFVIRGDEVRQIAPGQTGKIADYNYKLPDDFTGCVIAWNSNNLDSKVGGNYNYLYRLFRKSNLKPFERDISTEIYQAGSTPCNGVEGFNCYFPLQSYGFQPTNGVGYQPYRVVVLSFELLHAPATVCGPKKSTNLVKNKCVNFNFNGLTGTGVLTESNKKFLPFQQFGRDIADTTDAVRDPQTLEILDITPCSFGGVSVITPGTNTSNQVAVLYQDVNCTEVPVAIHADQLTPTWRVYSTGSNVFQTRAGCLIGAEHVNNSYECDIPIGAGICASYQTQTNSPRRARSVASQSIIAYTMSLGAENSVAYSNNSIAIPTNFTISVTTEILPVSMTKTSVDCTMYICGDSTECSNLLLQYGSFCTQLNRALTGIAVEQDKNTQEVFAQVKQIYKTPPIKDFGGFNFSQILPDPSKPSKRSFIEDLLFNKVTLADAGFIKQYGDCLGDIAARDLICAQKFNGLTVLPPLLTDEMIAQYTSALLAGTITSGWTFGAGAALQIPFAMQMAYRFNGIGVTQNVLYENQKLIANQFNSAIGKIQDSLSSTASALGKLQDVVNQNAQALNTLVKQLSSNFGAISSVLNDILSRLDPPEAEVQIDRLITGRLQSLQTYVTQQLIRAAEIRASANLAATKMSECVLGQSKRVDFCGKGYHLMSFPQSAPHGVVFLHVTYVPAQEKNFTTAPAICHDGKAHFPREGVFVSNGTHWFVTQRNFYEPQIITTDNTFVSGNCDVVIGIVNNTVYDPLQPELDSFKEELDKYFKNHTSPDVDLGDISGINASVVNIQKEIDRLNEVAKNLNESLIDLQELGKYEQYIKWPWYIWLGFIAGLIAIVMVTIMLCCMTSCCSCLKGCCSCGSCCKFDEDDSEPVLKGVKLHYT (SEQ ID NO: 14)
Последовательность ДНК или РНК S-(deg-RBD-122-165-234) штамма Wuhan S-2P (от 5'-конца к 3'-концу):
atgttcgtcttcctggtcctgctgcctctggtctcctcacagtgcgtcaatctgacaactcggactcagctgccacctgcttatactaatagcttcaccagaggcgtgtactatcctgacaaggtgtttagaagctccgtgctgcactctacacaggatctgtttctgccattctttagcaacgtgacctggttccacgccatccacgtgagcggcaccaatggcacaaagcggttcgacaatcccgtgctgccttttaacgatggcgtgtacttcgcctctaccgagaagagcaacatcatcagaggctggatctttggcaccacactggactccaagacacagtctctgctgatcgtgaaccaagccaccaacgtggtcatcaaggtgtgcgagttccagttttgtaatgatcccttcctgggcgtgtactatcacaagaacaataagagctggatggagtccgagtttagagtgtattctagcgccaaccagtgcacatttgagtacgtgagccagcctttcctgatggacctggagggcaagcagggcaatttcaagaacctgagggagttcgtgtttaagaatatcgacggctacttcaaaatctactctaagcacacccccatcaacctggtgcgcgacctgcctcagggcttcagcgccctggagcccctggtggatctgcctatcggcatccagatcacccggtttcagacactgctggccctgcacagaagctacctgacacccggcgactcctctagcggatggaccgccggcgctgccgcctactatgtgggctacctccagccccggaccttcctgctgaagtacaacgagaatggcaccatcacagacgcagtggattgcgccctggaccccctgagcgagacaaagtgtacactgaagtcctttaccgtggagaagggcatctatcagacatccaatttcagggtgcagccagccgaggcgatcgtgcgctttcctgaaatcacaaacctgtgcccatttggcgaggtgttcgaggcaacccgcttcgccagcgtgtacgcctggaataggaagcggatcagcaactgcgtggccgactatagcgtgctgtacaactccgcctctttcagcacctttaagtgctatggcgtgtcccccacaaagctgaatgacctgtgctttaccaacgtctacgccgattctttcgtgatcaggggcgacgaggtgcgccagatcgcccccggccagacaggcaagatcgcagactacaattataagctgccagacgatttcaccggctgcgtgatcgcctggaacagcaacaatctggattccaaagtgggcggcaactacaattatctgtaccggctgtttagaaagagcaatctgaagcccttcgagagggacatctctacagaaatctaccaggccggcagcaccccttgcaatggcgtggagggctttaactgttatttcccactccagtcctacggcttccagcccacaaacggcgtgggctatcagccttaccgcgtggtggtgctgagctttgagctgctgcacgccccagcaacagtgtgcggccccaagaagtccaccaatctggtgaagaacaagtgcgtgaacttcaacttcaacggcctgaccggcacaggcgtgctgaccgagtccaacaagaagttcctgccatttcagcagttcggcagggacatcgcagataccacagacgccgtgcgcgacccacagaccctggagatcctggacatcacaccctgctctttcggcggcgtgagcgtgatcacacccggcaccaatacaagcaaccaggtggccgtgctgtatcaggacgtgaattgtaccgaggtgcccgtggctatccacgccgatcagctgaccccaacatggcgggtgtacagcaccggctccaacgtcttccagacaagagccggatgcctgatcggagcagagcacgtgaacaattcctatgagtgcgacatcccaatcggcgccggcatctgtgcctcttaccagacccagacaaactctcccagaagagcccggagcgtggcctcccagtctatcatcgcctataccatgtccctgggcgccgagaacagcgtggcctactctaacaatagcatcgccatcccaaccaacttcacaatctctgtgaccacagagatcctgcccgtgtccatgaccaagacatctgtggactgcacaatgtatatctgtggcgattctaccgagtgcagcaacctgctgctccagtacggcagcttttgtacccagctgaatagagccctgacaggcatcgccgtggagcaggataagaacacacaggaggtgttcgcccaggtgaagcaaatctacaagaccccccctatcaaggactttggcggcttcaatttttcccagatcctgcctgatccatccaagccttctaagcggagctttatcgaggacctgctgttcaacaaggtgaccctggccgatgccggcttcatcaagcagtatggcgattgcctgggcgacatcgcagccagggacctgatctgcgcccagaagtttaatggcctgaccgtgctgccacccctgctgacagatgagatgatcgcacagtacacaagcgccctgctggccggcaccatcacatccggatggaccttcggcgcaggagccgccctccagatcccctttgccatgcagatggcctataggttcaacggcatcggcgtgacccagaatgtgctgtacgagaaccagaagctgatcgccaatcagtttaactccgccatcggcaagatccaggacagcctgtcctctacagccagcgccctgggcaagctccaggatgtggtgaatcagaacgcccaggccctgaataccctggtgaagcagctgagcagcaacttcggcgccatctc
ctccagacctatgtgacacagcagctgatcagggccgccgagatcagggccagcgccaatctggcagcaaccaagatgtccgagtgcgtgctgggccagtctaagagagtggacttttgtggcaagggctatcacctgatgtccttccctcagtctgccccacacggcgtggtgtttctgcacgtgacctacgtgcccgcccaggagaagaacttcaccacagcccctgccatctgccacgatggcaaggcccactttccaagggagggcgtgttcgtgtccaacggcacccactggtttgtgacacagcgcaatttctacgagccccagatcatcaccacagacaacaccttcgtgagcggcaactgtgacgtggtcatcggcatcgtgaacaataccgtgtatgatccactccagcccgagctggacagctttaaggaggagctggataagtatttcaagaatcacacctcccctgacgtggatctgggcgacatcagcggcatcaatgcctccgtggtgaacatccagaaggagatcgaccgcctgaacgaggtggctaagaatctgaacgagagcctgatcgacctccaggagctgggcaagtatgagcagtacatcaagtggccctggtacatctggctgggcttcatcgccggcctgatcgccatcgtgatggtgaccatcatgctgtgctgtatgacatcctgctgttcttgcctgaagggctgctgtagctgtggctcctgctgtaagtttgacgaggatgactctgaacctgtgctgaagggcgtgaagctgcattacacctaa(SEQ ID NO: 39)
Белковая последовательность S-(deg-RBD-122-165-234) для штамма Wuhan S-2P (от N-конца к C-концу):
MFVFLVLLPLVSSQCVNLTTRTQLPPAYTNSFTRGVYYPDKVFRSSVLHSTQDLFLPFFSNVTWFHAIHVSGTNGTKRFDNPVLPFNDGVYFASTEKSNIIRGWIFGTTLDSKTQSLLIVNQATNVVIKVCEFQFCNDPFLGVYYHKNNKSWMESEFRVYSSANQCTFEYVSQPFLMDLEGKQGNFKNLREFVFKNIDGYFKIYSKHTPINLVRDLPQGFSALEPLVDLPIGIQITRFQTLLALHRSYLTPGDSSSGWTAGAAAYYVGYLQPRTFLLKYNENGTITDAVDCALDPLSETKCTLKSFTVEKGIYQTSNFRVQPAEAIVRFPQITNLCPFGEVFQATRFASVYAWNRKRISNCVADYSVLYNSASFSTFKCYGVSPTKLNDLCFTNVYADSFVIRGDEVRQIAPGQTGKIADYNYKLPDDFTGCVIAWNSNNLDSKVGGNYNYLYRLFRKSNLKPFERDISTEIYQAGSTPCNGVEGFNCYFPLQSYGFQPTNGVGYQPYRVVVLSFELLHAPATVCGPKKSTNLVKNKCVNFNFNGLTGTGVLTESNKKFLPFQQFGRDIADTTDAVRDPQTLEILDITPCSFGGVSVITPGTNTSNQVAVLYQDVNCTEVPVAIHADQLTPTWRVYSTGSNVFQTRAGCLIGAEHVNNSYECDIPIGAGICASYQTQTNSPRRARSVASQSIIAYTMSLGAENSVAYSNNSIAIPTNFTISVTTEILPVSMTKTSVDCTMYICGDSTECSNLLLQYGSFCTQLNRALTGIAVEQDKNTQEVFAQVKQIYKTPPIKDFGGFNFSQILPDPSKPSKRSFIEDLLFNKVTLADAGFIKQYGDCLGDIAARDLICAQKFNGLTVLPPLLTDEMIAQYTSALLAGTITSGWTFGAGAALQIPFAMQMAYRFNGIGVTQNVLYENQKLIANQFNSAIGKIQDSLSSTASALGKLQDVV

RAAEIRASANLAATKMSECVLGQSKRVDFCGKGYHLMSFPQSAPHGVVFLHVTYVPAQEKNFTTAPAICHDGKAHFPREGVFVSNGTHWFVTQRNFYEPQIITTDNTFVSGNCDVVIGIVNNTVYDPLQPELDSFKEELDKYFKNHTSPDVDLGDISGINASVVNIQKEIDRLNEVAKNLNESLIDLQELGKYEQYIKWPWYIWLGFIAGLIAIVMVTIMLCCMTSCCSCLKGCCSCGSCCKFDEDDSEPVLKGVKLHYT(SEQ ID NO: 40)
Пример 5. Гликозилирование влияет на клеточный и цитокиновый ответ
Чтобы охарактеризовать ответ Т-клеток, спленоциты от иммунизированных мышей выделяли и инкубировали с пулом пептидов S-белка для измерения с помощью анализа Elispot Т-клеток, секретирующих гранзим B (GrzB). Было показано, что S-(deg-S2) и S-(S2-1194) индуцировали больше клеток, секретирующих GrzB, чем WT и S-(deg-RBD) после инкубации с полноразмерным WT S (фиг. 7A), RBD (фиг. 7B) и пептиды S2 (фиг. 7C), что свидетельствует о том, что гликозилирование S2 регулирует Т-клеточный ответ. Для дальнейшего изучения влияния на CD4+ и CD8+ Т-клетки, выделенные Т-клетки инкубировали с дендритными клетками костного мозга (DC) и пулом пептидов WTS для измерения с помощью проточной цитометрии Т-клеток, секретирующих IFNγ. Не было существенной разницы в количестве IFNγ-секретирующих CD4+ T-клеток среди всех вакцинированных мышей (фиг. 7D), но мРНК-вакцина S-(deg-S2) или S-(S2-1194) индуцировала больше IFNγ-секретирующие CD8+ Т-клетки, предполагая, что гликозилирование в S2 регулирует ответ CD8+ Т-клеток (фиг. 7E).
Для анализа экспрессии цитокинов среду от спленоцитов, инкубированных с пулом полноразмерных пептидов WTS, анализировали с помощью ELISA. Было показано, что спленоциты мышей, иммунизированных S-(deg-S2) и S-(S2-1194), секретировали более высокие уровни цитокинов T-хелпер-1 (TH1) (IFNγ, IL-2 и IL-12) (фиг. 8 A, B и E), тогда как спленоциты мышей, иммунизированных WT и S-(deg-RBD), секретировали более высокие уровни цитокинов Т-хелперов-2 (TH2) (IL-4, IL-6 и IL-13) (фиг. 8 C, D и F). В целом, все РНК-вакцины с мутацией сайтов гликозилирования индуцировали ответы антител и CD4+ и CD8+ Т-клеток и соответствующие цитокины с более сильным IFNγ-продуцирующим ответом CD8+ Т-клеток у мышей, иммунизированных S-(deg-S2) и S-(S2-1194). Эти результаты позволяют предположить, что гликозилирование S2 регулирует ответ Т-клеток и экспрессию цитокинов.
Пример 6. Дегликозилирование S2-индуцированного ответа развернутого белка
Чтобы исследовать, как гликозилирование на S2 влияет на иммунный ответ, клетки HEK293 трансфицировали плазмидой вариантов экспрессии S-белка, стабилизированной до слияния. Было показано, что S-(deg-S2) и S-(S2-1194) плохо экспрессировались, но уровни белков S-(deg-S2) и S-(S2-1194) в некоторой степени восстанавливались после обработки MG132, ингибитором протеасом (фиг. 9). Анализ трансляции in vitro показал, что мутация сайтов гликозилирования в последовательности мРНК не влияет на эффективность трансляции (фиг. 10). Эти результаты предполагают, что удаление гликозилирования из S2 вызывает деградацию транслируемого белка in vivo. Чтобы определить, приводит ли удаление гликозилирования на S2 к экспрессии неправильно свернутого или развернутого белка, плазматическую мембрану и эндоплазматический ретикулум (ER) изолировали для изучения распределения S-белка, поскольку неправильно свернутые или развернутые S-белки могли быть уложены в стопку в ER для рефолдинга или разрушиться в результате деградации, связанной с ER. После того, как клетки HEK293 были трансфицированы мРНК через 48 часов, все варианты белков S существовали в плазматической мембране, цитозоле и ER. Однако в плазматической мембране было больше белков WT и S-(deg-RBD), тогда как в ЭР было больше белков S-(deg-S2) и S-(S2-1194) (фиг. 11). Кроме того, клетки HEK293 были трансфицированы мРНК, которая кодировала растворимую версию S-(deg-S2) до слияния, и белок S-(S2-1194) не мог секретироваться в среду (фиг. 16), что позволяет предположить, что дегликозилирование в S2 повлияло на укладку S-белка. Поскольку повышенное содержание развернутого белка S в ER должно запускать ответ развернутого белка (UPR), маркерные белки UPR BiP/GRP78, XBP1 и p-eIF2α исследовали в клетках HEK293, трансфицированных РНК, через 48 часов. Результаты показали, что уровень BiP/GRP78 и XBP1 повышался, а уровень p-eIF2α был выше, чем в группах WT и S-(deg-RBD) в группах в клетках, трансфицированных S-(deg-S2) и S-(S2-1194) (фиг. 12). Кроме того, удаление гликозилирования на S2 индуцировало больше клеток апоптоза, чем WT и S-(deg-RBD) (фиг. 13), что свидетельствует о том, что дегликозилирование S2 индуцировало более высокий уровень стресса ER, чем WT и S-(deg-RBD) белок, а гликозилирование на S2 влияло на фолдинг белка, чтобы регулировать экспрессию UPR.
Пример 7. Гликозилирование S2 влияло на экспрессию MHC I на дендритных клетках (DC)
Чтобы изучить, приводит ли UPR к смещенному иммунному ответу, с помощью проточной цитометрии измеряли главный комплекс гистосовместимости класса I (MHC I) и класса II (MHC II) на DC, которые необходимы для презентации интернализованных молекул после процессинга. После того, как DC инкубировали с вариантами мРНК-вакцины, происходили активация МНС I/II среди всех вакцин, а мРНК-вакцина S-(deg-S2) или S-(S2-1194) индуцировала большее количество DC с экспрессией МНС I, чем WT и S-(deg-RBD) (фиг. 14 и фиг. 17), предполагая, что UPR регулирует экспрессию MHC I на DC.
Пример 8. Сайты гликозилирования в шиповидном белке влияют на стабильность S-белка и, следовательно, на ответ CD8+ Т-клеток
Чтобы изучить, какие сайты гликозилирования регулируют иммунный ответ хозяина, мы применяли вакцину S-(deg-RBD) в качестве модельной системы, поскольку она индуцировала такой же уровень антител, что и дикий тип, и обладала лучшей нейтрализующей активностью в отношении четырех рассматриваемых вариантов, чем дикий тип. В настоящем документе мы удалили сайты гликозилирования RBD и сайт гликозилирования N-801 (S-(deg-RBD-801)) или сайт гликозилирования N-1194 (S-(deg-RBD-1194)) в S2, поскольку эти сайты гликозилирования участвовали в экспрессии белка S, особенно сайт гликозилирования N-1194, который участвует в целостности S-белка и его аффинности связывания. Поскольку сайты гликозилирования N-122, N-165 и N-234 регулировали структуру RBD и влияли на нейтрализующую активность антител, мы удалили эти сайты гликозилирования для создания вакцины S-(deg-RBD-122-165-234) (фиг. 15). После того, как клетки HEK293 были трансфицированы экспрессионной плазмидой, стабилизированной до слияния вариантов S-белка, было показано, что S-(deg-RBD-801) и S-(deg-RBD-122-165-234) плохо экспрессируются, а S-(deg-RBD-1194) резко снижали экспрессию белка, но уровни этих вариантов белков восстанавливались до некоторой степени после обработки MG132, что свидетельствует о том, что вариант вакцины индуцировал деградацию транслируемого белка (фиг. 21А). После подтверждения экспрессии варианта белка S до слияния в форме мРНК-LNP в клеточной линии HEK293 (фиг. 21B) мышей иммунизировали различными вакцинами. Было показано, что S-(deg-RBD-801), S-(deg-RBD-1194) и S-(deg-RBD-122-165-234) индуцируют меньший титр IgG против полностью гликозилированного WT S (фиг. 18 A) и белка RBD (фиг. 18 B), но S-(deg-RBD-801) и S-(deg-RBD-122-165-234) имели в анализе ELISA более высокий титр IgG против дегликозилированного антигена RBD (фиг. 18 C) по сравнению с немодифицированной мРНК, что позволяет предположить, что гликозилирование этих сайтов гликозилирования регулирует продукцию антител. Для анализа нейтрализующей активности антител, полученных от иммунизированных мышей, анализ нейтрализации псевдовируса показал, что S-(deg-RBD-801), S-(deg-RBD-1194) и S-(deg-RBD-122-165-234) мРНК-вакцины генерировали антитела с пониженной нейтрализующей активностью против псевдовируса дикого типа (фиг. 19), но с более высокой нейтрализующей активностью в отношении четырех вызывающих озабоченность вариантов, по сравнению с диким типом (фиг. 19 и таблица 2).
Чтобы охарактеризовать Т-клеточный ответ, спленоциты иммунизированных мышей инкубировали с пулом пептидов белка S, RBD и S2, затем определяли гранзим B (GrzB)-секретирующие Т-клетки с помощью анализа ELISpot. Было показано, что S-(deg-RBD-801), S-(deg-RBD-1194) и S-(deg-RBD-122-165-234) индуцируют больше GrzB-секретирующих Т-клеток, чем WT, во всех пулах пептидов, в особенности, в S-(deg-RBD-1194) (фиг. 20 и фиг. 22А, 22В). После инкубации выделенных CD4+ и CD8+ Т-клеток с костномозговыми дендритными клетками и пулом пептидов WTS для измерения IFNγ-секретирующих Т-клеток с помощью проточной цитометрии не было выявлено существенной разницы в количестве IFNγ-секретирующих CD4+ Т-клеток среди всех вакцинированных мышей (фиг. 22C), но S-(deg-RBD-801), S-(deg-RBD-1194) и S-(deg-RBD-122-165-234) мРНК-вакцины индуцировали больше IFNγ-секретирующие CD8+ Т-клетки, что позволяет предположить, что эти вакцины индуцировали более сильный ответ CD8+ Т-клеток (фиг. 20). В целом, S-(deg-RBD-801), S-(deg-RBD-1194) и S-(deg-RBD-122-165-234) снижали уровень экспрессии белка в плазмидной системе и индуцировали меньше антител, но увеличил ответ CD8+ Т-клеток, особенно в S-(deg-RBD-1194). Эти результаты позволяют предположить, что гликозилирование сайтов гликозилирования, участвующих в укладке или стабилизации S, регулирует иммунный ответ хозяина.
Пример 9. Дизайн и синтез поли(дисульфидов) на основе гуанидина
Мы разработали серию полимеров и групп на основе гуанидина и/или цвиттерионных головок, прикрепленных к липидному хвосту, и исследовали их способность доставлять мРНК шиповидного белка. Как показано на фиг. 23, пропагатор P1, включающий гуанидиновые группы, и пропагатор мультивалентного дисплея P2 облегчают сцепление мРНК с полимерами за счет образования прочных солевых мостиков между гуанидинами и фосфатами в мРНК. Как только мРНК, инкапсулированная в полимер, достигает цитоплазмы, дисульфид линкеры могут расщепляться внутриклеточным глутатионом с высвобождением мРНК. Кроме того, деградированные дисульфидные мономеры также снижают цитотоксичность, предотвращая накопление высокомолекулярных полимеров внутри клетки.
Для получения полимеров моногуанидин-содержащий дисульфидный мономер синтезировали в соответствии с ранее опубликованными процедурами (Gasparini, G.; Bang, E.-K.; Molinard, G.; Tulumello, D. V.; Ward, S.; Kelley, S. O.; Roux, A.; Sakai, N.; Matile, S., J. Am. Chem. Soc. 2014, 136, 6069-6074), и мономер тригуанидиндисульфида синтезировали из линкера нитрилотриуксусной кислоты с получением настоящего мономера тримерного гуанидина. Пропагатор Pb, содержащий два напряженных дисульфида помимо гуанидиновой группы, был разработан для образования разветвленной конфигурации полимера. Пропагаторы P3 и Pz были сконструированы как спейсер, который может способствовать выходу захваченной молекулы из эндосомы.
На фиг. 24 показана сконструированная структура инициаторов (I0), пропагаторов (P1, P2) и процесс полимеризации/деполимеризации.
Полимеризацию P1, P2, P3 и Pb проводили в дегазированном водном растворе при комнатной температуре. Вкратце, в присутствии 5 мМ инициатора 1 и 200 мМ пропагатора Р в 1 М буфере TEOA с рН 7 энергично перемешивали в течение 30 минут. Терминацию проводили добавлением 0,5 М йодацетамида. Для скрининга оптимального полимера для эффективной внутриклеточной доставки мРНК проводили сополимеризацию различных пропагаторов и оценивали их способность к инкапсуляции и эффективность трансфекции с помощью кодирующей GFP мРНК в клетках HEK293T. Сополимер (P1/P3) и (P2/P3) готовили в соотношении 2:1, (P1/Pb) и (P2/Pb) готовили в соотношении 4:1.
Чтобы выбрать оптимальный полимер для лучшей инкапсуляции и эффективной внутриклеточной доставки мРНК, 8 типов синтезированных гомополимеров и гетеросополимеров исследовали на их способность инкапсулировать мРНК GFP. Как показано на фиг. 25А, все сополимеры, содержащие гуанидиновые группы, были способны ингибировать миграцию мРНК GFP с отношением N/P = 10. В особенности, сополимер P2/P3 и P2/Pb с тригуанидиновым фрагментом приводил к более высокой способности образовывать комплексы с мРНК, которая была сравнима с результатом, опосредованным традиционно используемым агентом трансфекции полиэтиленимином (PEI). Средний размер полученного нанокластерного комплекса P2/P3-мРНК составлял примерно 90 нм с помощью ПЭМ (фиг. 25В). Кроме того, разветвленные полимеры P1/Pb и P2/Pb также показали сходную способность инкапсулировать мРНК. Разветвленные гуанидинии привлекли внимание своей хорошей доступностью и гибкостью для функциональных материалов.
Затем мы оценили эффективность трансфекции GFP-мРНК в клетках HEK293T с применением различных сополимеров. Во-первых, мы обнаружили, что сополимер P1/P3 проявляет хорошую способность к трансфекции мРНК при N/P = 10, что более эффективно, чем P1 или P3 по отдельности и PEI (фиг. 26A). Результат показал, что полимер P1 или P3 не подходит для внутриклеточной доставки, в то время как бифункциональный сополимер P1/P3 в значительной степени трансфицировал мРНК. Случайное расположение гуанидиновой группы путем сополимеризации со спейсером из диэтилентриамина может быть подходящим для соответствия фосфатному заряду нуклеотида. Затем мы изучили влияние различных сополимеров на активность трансфекции. На фиг. 26B комбинация P2/P3 показала повышенную экспрессию GFP, что указывает на то, что тригуанидиновая группа усиливала инкапсуляцию мРНК, а также могла высвобождать мРНК после доставки в цитоплазму. Однако разветвленные поли(дисульфиды) P1/Pb и P2/Pb не обнаруживали удовлетворительного высвобождения грузов мРНК, хотя полное комплексообразование с мРНК осуществлялось с применением P2/Pb (фиг. 26A). Мы дополнительно оптимизировали соотношение N/P (1, 5, 10 и 20), применяя выдающийся сополимер P2/P3. Обнаружено, что наилучшая эффективность для трансфекции составляет 10 (фиг. 26C).
На основании результатов мРНК GFP была получена мРНК шиповидного белка дикого типа, которая была инкапсулирована с помощью полиГу при различных соотношениях N/P (фиг. 27). ПолиГу продемонстрировал хорошую способность нейтрализовать заряд мРНК шиповидного белка и успешно захватил его при соотношении N/P, равном 1.
Затем мы трансфицировали мРНК шиповидного белка в клетки HEK293T и провели вестерн-блоттинг. Клетки HEK293T трансфицировали 3 мкг мРНК шиповидного белка. Через 48 часов после трансфекции клетки анализировали на экспрессию шиповидных белков с помощью вестерн-блоттинга с применением специфичных для шиповидных белков антител. Результат показал значительную полосу шиповидного белка SARS-Cov-2 примерно 250 кДа, и буфер PBS с мРНК шиповидного белка был применен в качестве отрицательного контроля (фиг. 28), что подтвердило возможность применения полигуанина в качестве наноносителя для трансфекции мРНК in vitro. Кроме того, полиГу не проявлял явной цитотоксичности до 10 мкг (50 мкг/мл) (фиг. 29). В то время как липидные наночастицы (LNP) проявляли гораздо более высокую токсичность по отношению к клеткам с высокой нагрузкой комплексов.
В заключение, мы разработали серию поли(дисульфидов) и продемонстрировали, что комбинация гуанидильной группы и цвиттер-ионного спейсера демонстрирует высокую эффективность доставки мРНК in vitro. Эффективная внутриклеточная доставка через тиол-опосредованный путь поглощения штаммами дисульфидов расщепляется внутриклеточным глутатионом. Кроме того, деградация полимеров также минимизирует цитотоксичность по сравнению с обычно применяемыми LNP против SARS-Cov-2.
Все публикации и патентные заявки, цитируемые в данном описании, включены сюда в качестве отсылки, как если бы каждая отдельная публикация или патентная заявка были конкретно и отдельно указаны для включения в качестве отсылки.
Хотя вышеизложенное изобретение было описано довольно подробно посредством иллюстрации и примера в целях ясности понимания, специалистам в данной области техники будет очевидно, что в свете положений настоящего изобретения в него могут быть внесены определенные изменения и модификации без отклонения от сущности или объема прилагаемой формулы изобретения.
--->
СПИСОК ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> ACADEMIA SINICA
WONG, CHI-HUEY
WU, CHUNG-YI
MA, CHE
FAN, CHEN-YU
<120> MESSENGER RNA VACCINES AGAINST WIDE SPECTRUM OF CORONAVIRUS
VARIANTS
<130> G4590-15000PCT
<150> US 63/173,752
<151> 2021-04-12
<150> US 63/264,737
<151> 2021-12-01
<160> 52
<170> PatentIn version 3.5
<210> 1
<211> 3822
<212> DNA
<213> SARS-CoV-2 (Wuhan strain)
<400> 1
atgttcgtct tcctggtcct gctgcctctg gtctcctcac agtgcgtcaa tctgacaact 60
cggactcagc tgccacctgc ttatactaat agcttcacca gaggcgtgta ctatcctgac 120
aaggtgttta gaagctccgt gctgcactct acacaggatc tgtttctgcc attctttagc 180
aacgtgacct ggttccacgc catccacgtg agcggcacca atggcacaaa gcggttcgac 240
aatcccgtgc tgccttttaa cgatggcgtg tacttcgcct ctaccgagaa gagcaacatc 300
atcagaggct ggatctttgg caccacactg gactccaaga cacagtctct gctgatcgtg 360
aacaatgcca ccaacgtggt catcaaggtg tgcgagttcc agttttgtaa tgatcccttc 420
ctgggcgtgt actatcacaa gaacaataag agctggatgg agtccgagtt tagagtgtat 480
tctagcgcca acaactgcac atttgagtac gtgagccagc ctttcctgat ggacctggag 540
ggcaagcagg gcaatttcaa gaacctgagg gagttcgtgt ttaagaatat cgacggctac 600
ttcaaaatct actctaagca cacccccatc aacctggtgc gcgacctgcc tcagggcttc 660
agcgccctgg agcccctggt ggatctgcct atcggcatca acatcacccg gtttcagaca 720
ctgctggccc tgcacagaag ctacctgaca cccggcgact cctctagcgg atggaccgcc 780
ggcgctgccg cctactatgt gggctacctc cagccccgga ccttcctgct gaagtacaac 840
gagaatggca ccatcacaga cgcagtggat tgcgccctgg accccctgag cgagacaaag 900
tgtacactga agtcctttac cgtggagaag ggcatctatc agacatccaa tttcagggtg 960
cagccaaccg agtctatcgt gcgctttcct aatatcacaa acctgtgccc atttggcgag 1020
gtgttcaacg caacccgctt cgccagcgtg tacgcctgga ataggaagcg gatcagcaac 1080
tgcgtggccg actatagcgt gctgtacaac tccgcctctt tcagcacctt taagtgctat 1140
ggcgtgtccc ccacaaagct gaatgacctg tgctttacca acgtctacgc cgattctttc 1200
gtgatcaggg gcgacgaggt gcgccagatc gcccccggcc agacaggcaa gatcgcagac 1260
tacaattata agctgccaga cgatttcacc ggctgcgtga tcgcctggaa cagcaacaat 1320
ctggattcca aagtgggcgg caactacaat tatctgtacc ggctgtttag aaagagcaat 1380
ctgaagccct tcgagaggga catctctaca gaaatctacc aggccggcag caccccttgc 1440
aatggcgtgg agggctttaa ctgttatttc ccactccagt cctacggctt ccagcccaca 1500
aacggcgtgg gctatcagcc ttaccgcgtg gtggtgctga gctttgagct gctgcacgcc 1560
ccagcaacag tgtgcggccc caagaagtcc accaatctgg tgaagaacaa gtgcgtgaac 1620
ttcaacttca acggcctgac cggcacaggc gtgctgaccg agtccaacaa gaagttcctg 1680
ccatttcagc agttcggcag ggacatcgca gataccacag acgccgtgcg cgacccacag 1740
accctggaga tcctggacat cacaccctgc tctttcggcg gcgtgagcgt gatcacaccc 1800
ggcaccaata caagcaacca ggtggccgtg ctgtatcagg acgtgaattg taccgaggtg 1860
cccgtggcta tccacgccga tcagctgacc ccaacatggc gggtgtacag caccggctcc 1920
aacgtcttcc agacaagagc cggatgcctg atcggagcag agcacgtgaa caattcctat 1980
gagtgcgaca tcccaatcgg cgccggcatc tgtgcctctt accagaccca gacaaactct 2040
cccagaagag cccggagcgt ggcctcccag tctatcatcg cctataccat gtccctgggc 2100
gccgagaaca gcgtggccta ctctaacaat agcatcgcca tcccaaccaa cttcacaatc 2160
tctgtgacca cagagatcct gcccgtgtcc atgaccaaga catctgtgga ctgcacaatg 2220
tatatctgtg gcgattctac cgagtgcagc aacctgctgc tccagtacgg cagcttttgt 2280
acccagctga atagagccct gacaggcatc gccgtggagc aggataagaa cacacaggag 2340
gtgttcgccc aggtgaagca aatctacaag acccccccta tcaaggactt tggcggcttc 2400
aatttttccc agatcctgcc tgatccatcc aagccttcta agcggagctt tatcgaggac 2460
ctgctgttca acaaggtgac cctggccgat gccggcttca tcaagcagta tggcgattgc 2520
ctgggcgaca tcgcagccag ggacctgatc tgcgcccaga agtttaatgg cctgaccgtg 2580
ctgccacccc tgctgacaga tgagatgatc gcacagtaca caagcgccct gctggccggc 2640
accatcacat ccggatggac cttcggcgca ggagccgccc tccagatccc ctttgccatg 2700
cagatggcct ataggttcaa cggcatcggc gtgacccaga atgtgctgta cgagaaccag 2760
aagctgatcg ccaatcagtt taactccgcc atcggcaaga tccaggacag cctgtcctct 2820
acagccagcg ccctgggcaa gctccaggat gtggtgaatc agaacgccca ggccctgaat 2880
accctggtga agcagctgag cagcaacttc ggcgccatct ctagcgtgct gaatgacatc 2940
ctgagccggc tggacaaggt ggaggcagag gtgcagatcg accggctgat caccggccgg 3000
ctccagagcc tccagaccta tgtgacacag cagctgatca gggccgccga gatcagggcc 3060
agcgccaatc tggcagcaac caagatgtcc gagtgcgtgc tgggccagtc taagagagtg 3120
gacttttgtg gcaagggcta tcacctgatg tccttccctc agtctgcccc acacggcgtg 3180
gtgtttctgc acgtgaccta cgtgcccgcc caggagaaga acttcaccac agcccctgcc 3240
atctgccacg atggcaaggc ccactttcca agggagggcg tgttcgtgtc caacggcacc 3300
cactggtttg tgacacagcg caatttctac gagccccaga tcatcaccac agacaacacc 3360
ttcgtgagcg gcaactgtga cgtggtcatc ggcatcgtga acaataccgt gtatgatcca 3420
ctccagcccg agctggacag ctttaaggag gagctggata agtatttcaa gaatcacacc 3480
tcccctgacg tggatctggg cgacatcagc ggcatcaatg cctccgtggt gaacatccag 3540
aaggagatcg accgcctgaa cgaggtggct aagaatctga acgagagcct gatcgacctc 3600
caggagctgg gcaagtatga gcagtacatc aagtggccct ggtacatctg gctgggcttc 3660
atcgccggcc tgatcgccat cgtgatggtg accatcatgc tgtgctgtat gacatcctgc 3720
tgttcttgcc tgaagggctg ctgtagctgt ggctcctgct gtaagtttga cgaggatgac 3780
tctgaacctg tgctgaaggg cgtgaagctg cattacacct aa 3822
<210> 2
<211> 1273
<212> PRT
<213> SARS-CoV-2 (Wuhan strain)
<400> 2
Met Phe Val Phe Leu Val Leu Leu Pro Leu Val Ser Ser Gln Cys Val
1 5 10 15
Asn Leu Thr Thr Arg Thr Gln Leu Pro Pro Ala Tyr Thr Asn Ser Phe
20 25 30
Thr Arg Gly Val Tyr Tyr Pro Asp Lys Val Phe Arg Ser Ser Val Leu
35 40 45
His Ser Thr Gln Asp Leu Phe Leu Pro Phe Phe Ser Asn Val Thr Trp
50 55 60
Phe His Ala Ile His Val Ser Gly Thr Asn Gly Thr Lys Arg Phe Asp
65 70 75 80
Asn Pro Val Leu Pro Phe Asn Asp Gly Val Tyr Phe Ala Ser Thr Glu
85 90 95
Lys Ser Asn Ile Ile Arg Gly Trp Ile Phe Gly Thr Thr Leu Asp Ser
100 105 110
Lys Thr Gln Ser Leu Leu Ile Val Asn Asn Ala Thr Asn Val Val Ile
115 120 125
Lys Val Cys Glu Phe Gln Phe Cys Asn Asp Pro Phe Leu Gly Val Tyr
130 135 140
Tyr His Lys Asn Asn Lys Ser Trp Met Glu Ser Glu Phe Arg Val Tyr
145 150 155 160
Ser Ser Ala Asn Asn Cys Thr Phe Glu Tyr Val Ser Gln Pro Phe Leu
165 170 175
Met Asp Leu Glu Gly Lys Gln Gly Asn Phe Lys Asn Leu Arg Glu Phe
180 185 190
Val Phe Lys Asn Ile Asp Gly Tyr Phe Lys Ile Tyr Ser Lys His Thr
195 200 205
Pro Ile Asn Leu Val Arg Asp Leu Pro Gln Gly Phe Ser Ala Leu Glu
210 215 220
Pro Leu Val Asp Leu Pro Ile Gly Ile Asn Ile Thr Arg Phe Gln Thr
225 230 235 240
Leu Leu Ala Leu His Arg Ser Tyr Leu Thr Pro Gly Asp Ser Ser Ser
245 250 255
Gly Trp Thr Ala Gly Ala Ala Ala Tyr Tyr Val Gly Tyr Leu Gln Pro
260 265 270
Arg Thr Phe Leu Leu Lys Tyr Asn Glu Asn Gly Thr Ile Thr Asp Ala
275 280 285
Val Asp Cys Ala Leu Asp Pro Leu Ser Glu Thr Lys Cys Thr Leu Lys
290 295 300
Ser Phe Thr Val Glu Lys Gly Ile Tyr Gln Thr Ser Asn Phe Arg Val
305 310 315 320
Gln Pro Thr Glu Ser Ile Val Arg Phe Pro Asn Ile Thr Asn Leu Cys
325 330 335
Pro Phe Gly Glu Val Phe Asn Ala Thr Arg Phe Ala Ser Val Tyr Ala
340 345 350
Trp Asn Arg Lys Arg Ile Ser Asn Cys Val Ala Asp Tyr Ser Val Leu
355 360 365
Tyr Asn Ser Ala Ser Phe Ser Thr Phe Lys Cys Tyr Gly Val Ser Pro
370 375 380
Thr Lys Leu Asn Asp Leu Cys Phe Thr Asn Val Tyr Ala Asp Ser Phe
385 390 395 400
Val Ile Arg Gly Asp Glu Val Arg Gln Ile Ala Pro Gly Gln Thr Gly
405 410 415
Lys Ile Ala Asp Tyr Asn Tyr Lys Leu Pro Asp Asp Phe Thr Gly Cys
420 425 430
Val Ile Ala Trp Asn Ser Asn Asn Leu Asp Ser Lys Val Gly Gly Asn
435 440 445
Tyr Asn Tyr Leu Tyr Arg Leu Phe Arg Lys Ser Asn Leu Lys Pro Phe
450 455 460
Glu Arg Asp Ile Ser Thr Glu Ile Tyr Gln Ala Gly Ser Thr Pro Cys
465 470 475 480
Asn Gly Val Glu Gly Phe Asn Cys Tyr Phe Pro Leu Gln Ser Tyr Gly
485 490 495
Phe Gln Pro Thr Asn Gly Val Gly Tyr Gln Pro Tyr Arg Val Val Val
500 505 510
Leu Ser Phe Glu Leu Leu His Ala Pro Ala Thr Val Cys Gly Pro Lys
515 520 525
Lys Ser Thr Asn Leu Val Lys Asn Lys Cys Val Asn Phe Asn Phe Asn
530 535 540
Gly Leu Thr Gly Thr Gly Val Leu Thr Glu Ser Asn Lys Lys Phe Leu
545 550 555 560
Pro Phe Gln Gln Phe Gly Arg Asp Ile Ala Asp Thr Thr Asp Ala Val
565 570 575
Arg Asp Pro Gln Thr Leu Glu Ile Leu Asp Ile Thr Pro Cys Ser Phe
580 585 590
Gly Gly Val Ser Val Ile Thr Pro Gly Thr Asn Thr Ser Asn Gln Val
595 600 605
Ala Val Leu Tyr Gln Asp Val Asn Cys Thr Glu Val Pro Val Ala Ile
610 615 620
His Ala Asp Gln Leu Thr Pro Thr Trp Arg Val Tyr Ser Thr Gly Ser
625 630 635 640
Asn Val Phe Gln Thr Arg Ala Gly Cys Leu Ile Gly Ala Glu His Val
645 650 655
Asn Asn Ser Tyr Glu Cys Asp Ile Pro Ile Gly Ala Gly Ile Cys Ala
660 665 670
Ser Tyr Gln Thr Gln Thr Asn Ser Pro Arg Arg Ala Arg Ser Val Ala
675 680 685
Ser Gln Ser Ile Ile Ala Tyr Thr Met Ser Leu Gly Ala Glu Asn Ser
690 695 700
Val Ala Tyr Ser Asn Asn Ser Ile Ala Ile Pro Thr Asn Phe Thr Ile
705 710 715 720
Ser Val Thr Thr Glu Ile Leu Pro Val Ser Met Thr Lys Thr Ser Val
725 730 735
Asp Cys Thr Met Tyr Ile Cys Gly Asp Ser Thr Glu Cys Ser Asn Leu
740 745 750
Leu Leu Gln Tyr Gly Ser Phe Cys Thr Gln Leu Asn Arg Ala Leu Thr
755 760 765
Gly Ile Ala Val Glu Gln Asp Lys Asn Thr Gln Glu Val Phe Ala Gln
770 775 780
Val Lys Gln Ile Tyr Lys Thr Pro Pro Ile Lys Asp Phe Gly Gly Phe
785 790 795 800
Asn Phe Ser Gln Ile Leu Pro Asp Pro Ser Lys Pro Ser Lys Arg Ser
805 810 815
Phe Ile Glu Asp Leu Leu Phe Asn Lys Val Thr Leu Ala Asp Ala Gly
820 825 830
Phe Ile Lys Gln Tyr Gly Asp Cys Leu Gly Asp Ile Ala Ala Arg Asp
835 840 845
Leu Ile Cys Ala Gln Lys Phe Asn Gly Leu Thr Val Leu Pro Pro Leu
850 855 860
Leu Thr Asp Glu Met Ile Ala Gln Tyr Thr Ser Ala Leu Leu Ala Gly
865 870 875 880
Thr Ile Thr Ser Gly Trp Thr Phe Gly Ala Gly Ala Ala Leu Gln Ile
885 890 895
Pro Phe Ala Met Gln Met Ala Tyr Arg Phe Asn Gly Ile Gly Val Thr
900 905 910
Gln Asn Val Leu Tyr Glu Asn Gln Lys Leu Ile Ala Asn Gln Phe Asn
915 920 925
Ser Ala Ile Gly Lys Ile Gln Asp Ser Leu Ser Ser Thr Ala Ser Ala
930 935 940
Leu Gly Lys Leu Gln Asp Val Val Asn Gln Asn Ala Gln Ala Leu Asn
945 950 955 960
Thr Leu Val Lys Gln Leu Ser Ser Asn Phe Gly Ala Ile Ser Ser Val
965 970 975
Leu Asn Asp Ile Leu Ser Arg Leu Asp Pro Pro Glu Ala Glu Val Gln
980 985 990
Ile Asp Arg Leu Ile Thr Gly Arg Leu Gln Ser Leu Gln Thr Tyr Val
995 1000 1005
Thr Gln Gln Leu Ile Arg Ala Ala Glu Ile Arg Ala Ser Ala Asn
1010 1015 1020
Leu Ala Ala Thr Lys Met Ser Glu Cys Val Leu Gly Gln Ser Lys
1025 1030 1035
Arg Val Asp Phe Cys Gly Lys Gly Tyr His Leu Met Ser Phe Pro
1040 1045 1050
Gln Ser Ala Pro His Gly Val Val Phe Leu His Val Thr Tyr Val
1055 1060 1065
Pro Ala Gln Glu Lys Asn Phe Thr Thr Ala Pro Ala Ile Cys His
1070 1075 1080
Asp Gly Lys Ala His Phe Pro Arg Glu Gly Val Phe Val Ser Asn
1085 1090 1095
Gly Thr His Trp Phe Val Thr Gln Arg Asn Phe Tyr Glu Pro Gln
1100 1105 1110
Ile Ile Thr Thr Asp Asn Thr Phe Val Ser Gly Asn Cys Asp Val
1115 1120 1125
Val Ile Gly Ile Val Asn Asn Thr Val Tyr Asp Pro Leu Gln Pro
1130 1135 1140
Glu Leu Asp Ser Phe Lys Glu Glu Leu Asp Lys Tyr Phe Lys Asn
1145 1150 1155
His Thr Ser Pro Asp Val Asp Leu Gly Asp Ile Ser Gly Ile Asn
1160 1165 1170
Ala Ser Val Val Asn Ile Gln Lys Glu Ile Asp Arg Leu Asn Glu
1175 1180 1185
Val Ala Lys Asn Leu Asn Glu Ser Leu Ile Asp Leu Gln Glu Leu
1190 1195 1200
Gly Lys Tyr Glu Gln Tyr Ile Lys Trp Pro Trp Tyr Ile Trp Leu
1205 1210 1215
Gly Phe Ile Ala Gly Leu Ile Ala Ile Val Met Val Thr Ile Met
1220 1225 1230
Leu Cys Cys Met Thr Ser Cys Cys Ser Cys Leu Lys Gly Cys Cys
1235 1240 1245
Ser Cys Gly Ser Cys Cys Lys Phe Asp Glu Asp Asp Ser Glu Pro
1250 1255 1260
Val Leu Lys Gly Val Lys Leu His Tyr Thr
1265 1270
<210> 3
<211> 3822
<212> DNA
<213> Artificial Sequence
<220>
<223> S-(deg-RBD)
<400> 3
atgttcgtct tcctggtcct gctgcctctg gtctcctcac agtgcgtcaa tctgacaact 60
cggactcagc tgccacctgc ttatactaat agcttcacca gaggcgtgta ctatcctgac 120
aaggtgttta gaagctccgt gctgcactct acacaggatc tgtttctgcc attctttagc 180
aacgtgacct ggttccacgc catccacgtg agcggcacca atggcacaaa gcggttcgac 240
aatcccgtgc tgccttttaa cgatggcgtg tacttcgcct ctaccgagaa gagcaacatc 300
atcagaggct ggatctttgg caccacactg gactccaaga cacagtctct gctgatcgtg 360
aacaatgcca ccaacgtggt catcaaggtg tgcgagttcc agttttgtaa tgatcccttc 420
ctgggcgtgt actatcacaa gaacaataag agctggatgg agtccgagtt tagagtgtat 480
tctagcgcca acaactgcac atttgagtac gtgagccagc ctttcctgat ggacctggag 540
ggcaagcagg gcaatttcaa gaacctgagg gagttcgtgt ttaagaatat cgacggctac 600
ttcaaaatct actctaagca cacccccatc aacctggtgc gcgacctgcc tcagggcttc 660
agcgccctgg agcccctggt ggatctgcct atcggcatca acatcacccg gtttcagaca 720
ctgctggccc tgcacagaag ctacctgaca cccggcgact cctctagcgg atggaccgcc 780
ggcgctgccg cctactatgt gggctacctc cagccccgga ccttcctgct gaagtacaac 840
gagaatggca ccatcacaga cgcagtggat tgcgccctgg accccctgag cgagacaaag 900
tgtacactga agtcctttac cgtggagaag ggcatctatc agacatccaa tttcagggtg 960
cagccagccg aggcgatcgt gcgctttcct gaaatcacaa acctgtgccc atttggcgag 1020
gtgttcgagg caacccgctt cgccagcgtg tacgcctgga ataggaagcg gatcagcaac 1080
tgcgtggccg actatagcgt gctgtacaac tccgcctctt tcagcacctt taagtgctat 1140
ggcgtgtccc ccacaaagct gaatgacctg tgctttacca acgtctacgc cgattctttc 1200
gtgatcaggg gcgacgaggt gcgccagatc gcccccggcc agacaggcaa gatcgcagac 1260
tacaattata agctgccaga cgatttcacc ggctgcgtga tcgcctggaa cagcaacaat 1320
ctggattcca aagtgggcgg caactacaat tatctgtacc ggctgtttag aaagagcaat 1380
ctgaagccct tcgagaggga catctctaca gaaatctacc aggccggcag caccccttgc 1440
aatggcgtgg agggctttaa ctgttatttc ccactccagt cctacggctt ccagcccaca 1500
aacggcgtgg gctatcagcc ttaccgcgtg gtggtgctga gctttgagct gctgcacgcc 1560
ccagcaacag tgtgcggccc caagaagtcc accaatctgg tgaagaacaa gtgcgtgaac 1620
ttcaacttca acggcctgac cggcacaggc gtgctgaccg agtccaacaa gaagttcctg 1680
ccatttcagc agttcggcag ggacatcgca gataccacag acgccgtgcg cgacccacag 1740
accctggaga tcctggacat cacaccctgc tctttcggcg gcgtgagcgt gatcacaccc 1800
ggcaccaata caagcaacca ggtggccgtg ctgtatcagg acgtgaattg taccgaggtg 1860
cccgtggcta tccacgccga tcagctgacc ccaacatggc gggtgtacag caccggctcc 1920
aacgtcttcc agacaagagc cggatgcctg atcggagcag agcacgtgaa caattcctat 1980
gagtgcgaca tcccaatcgg cgccggcatc tgtgcctctt accagaccca gacaaactct 2040
cccagaagag cccggagcgt ggcctcccag tctatcatcg cctataccat gtccctgggc 2100
gccgagaaca gcgtggccta ctctaacaat agcatcgcca tcccaaccaa cttcacaatc 2160
tctgtgacca cagagatcct gcccgtgtcc atgaccaaga catctgtgga ctgcacaatg 2220
tatatctgtg gcgattctac cgagtgcagc aacctgctgc tccagtacgg cagcttttgt 2280
acccagctga atagagccct gacaggcatc gccgtggagc aggataagaa cacacaggag 2340
gtgttcgccc aggtgaagca aatctacaag acccccccta tcaaggactt tggcggcttc 2400
aatttttccc agatcctgcc tgatccatcc aagccttcta agcggagctt tatcgaggac 2460
ctgctgttca acaaggtgac cctggccgat gccggcttca tcaagcagta tggcgattgc 2520
ctgggcgaca tcgcagccag ggacctgatc tgcgcccaga agtttaatgg cctgaccgtg 2580
ctgccacccc tgctgacaga tgagatgatc gcacagtaca caagcgccct gctggccggc 2640
accatcacat ccggatggac cttcggcgca ggagccgccc tccagatccc ctttgccatg 2700
cagatggcct ataggttcaa cggcatcggc gtgacccaga atgtgctgta cgagaaccag 2760
aagctgatcg ccaatcagtt taactccgcc atcggcaaga tccaggacag cctgtcctct 2820
acagccagcg ccctgggcaa gctccaggat gtggtgaatc agaacgccca ggccctgaat 2880
accctggtga agcagctgag cagcaacttc ggcgccatct ctagcgtgct gaatgacatc 2940
ctgagccggc tggacaaggt ggaggcagag gtgcagatcg accggctgat caccggccgg 3000
ctccagagcc tccagaccta tgtgacacag cagctgatca gggccgccga gatcagggcc 3060
agcgccaatc tggcagcaac caagatgtcc gagtgcgtgc tgggccagtc taagagagtg 3120
gacttttgtg gcaagggcta tcacctgatg tccttccctc agtctgcccc acacggcgtg 3180
gtgtttctgc acgtgaccta cgtgcccgcc caggagaaga acttcaccac agcccctgcc 3240
atctgccacg atggcaaggc ccactttcca agggagggcg tgttcgtgtc caacggcacc 3300
cactggtttg tgacacagcg caatttctac gagccccaga tcatcaccac agacaacacc 3360
ttcgtgagcg gcaactgtga cgtggtcatc ggcatcgtga acaataccgt gtatgatcca 3420
ctccagcccg agctggacag ctttaaggag gagctggata agtatttcaa gaatcacacc 3480
tcccctgacg tggatctggg cgacatcagc ggcatcaatg cctccgtggt gaacatccag 3540
aaggagatcg accgcctgaa cgaggtggct aagaatctga acgagagcct gatcgacctc 3600
caggagctgg gcaagtatga gcagtacatc aagtggccct ggtacatctg gctgggcttc 3660
atcgccggcc tgatcgccat cgtgatggtg accatcatgc tgtgctgtat gacatcctgc 3720
tgttcttgcc tgaagggctg ctgtagctgt ggctcctgct gtaagtttga cgaggatgac 3780
tctgaacctg tgctgaaggg cgtgaagctg cattacacct aa 3822
<210> 4
<211> 1273
<212> PRT
<213> Artificial Sequence
<220>
<223> S-(deg-RBD)
<400> 4
Met Phe Val Phe Leu Val Leu Leu Pro Leu Val Ser Ser Gln Cys Val
1 5 10 15
Asn Leu Thr Thr Arg Thr Gln Leu Pro Pro Ala Tyr Thr Asn Ser Phe
20 25 30
Thr Arg Gly Val Tyr Tyr Pro Asp Lys Val Phe Arg Ser Ser Val Leu
35 40 45
His Ser Thr Gln Asp Leu Phe Leu Pro Phe Phe Ser Asn Val Thr Trp
50 55 60
Phe His Ala Ile His Val Ser Gly Thr Asn Gly Thr Lys Arg Phe Asp
65 70 75 80
Asn Pro Val Leu Pro Phe Asn Asp Gly Val Tyr Phe Ala Ser Thr Glu
85 90 95
Lys Ser Asn Ile Ile Arg Gly Trp Ile Phe Gly Thr Thr Leu Asp Ser
100 105 110
Lys Thr Gln Ser Leu Leu Ile Val Asn Asn Ala Thr Asn Val Val Ile
115 120 125
Lys Val Cys Glu Phe Gln Phe Cys Asn Asp Pro Phe Leu Gly Val Tyr
130 135 140
Tyr His Lys Asn Asn Lys Ser Trp Met Glu Ser Glu Phe Arg Val Tyr
145 150 155 160
Ser Ser Ala Asn Asn Cys Thr Phe Glu Tyr Val Ser Gln Pro Phe Leu
165 170 175
Met Asp Leu Glu Gly Lys Gln Gly Asn Phe Lys Asn Leu Arg Glu Phe
180 185 190
Val Phe Lys Asn Ile Asp Gly Tyr Phe Lys Ile Tyr Ser Lys His Thr
195 200 205
Pro Ile Asn Leu Val Arg Asp Leu Pro Gln Gly Phe Ser Ala Leu Glu
210 215 220
Pro Leu Val Asp Leu Pro Ile Gly Ile Asn Ile Thr Arg Phe Gln Thr
225 230 235 240
Leu Leu Ala Leu His Arg Ser Tyr Leu Thr Pro Gly Asp Ser Ser Ser
245 250 255
Gly Trp Thr Ala Gly Ala Ala Ala Tyr Tyr Val Gly Tyr Leu Gln Pro
260 265 270
Arg Thr Phe Leu Leu Lys Tyr Asn Glu Asn Gly Thr Ile Thr Asp Ala
275 280 285
Val Asp Cys Ala Leu Asp Pro Leu Ser Glu Thr Lys Cys Thr Leu Lys
290 295 300
Ser Phe Thr Val Glu Lys Gly Ile Tyr Gln Thr Ser Asn Phe Arg Val
305 310 315 320
Gln Pro Ala Glu Ala Ile Val Arg Phe Pro Gln Ile Thr Asn Leu Cys
325 330 335
Pro Phe Gly Glu Val Phe Gln Ala Thr Arg Phe Ala Ser Val Tyr Ala
340 345 350
Trp Asn Arg Lys Arg Ile Ser Asn Cys Val Ala Asp Tyr Ser Val Leu
355 360 365
Tyr Asn Ser Ala Ser Phe Ser Thr Phe Lys Cys Tyr Gly Val Ser Pro
370 375 380
Thr Lys Leu Asn Asp Leu Cys Phe Thr Asn Val Tyr Ala Asp Ser Phe
385 390 395 400
Val Ile Arg Gly Asp Glu Val Arg Gln Ile Ala Pro Gly Gln Thr Gly
405 410 415
Lys Ile Ala Asp Tyr Asn Tyr Lys Leu Pro Asp Asp Phe Thr Gly Cys
420 425 430
Val Ile Ala Trp Asn Ser Asn Asn Leu Asp Ser Lys Val Gly Gly Asn
435 440 445
Tyr Asn Tyr Leu Tyr Arg Leu Phe Arg Lys Ser Asn Leu Lys Pro Phe
450 455 460
Glu Arg Asp Ile Ser Thr Glu Ile Tyr Gln Ala Gly Ser Thr Pro Cys
465 470 475 480
Asn Gly Val Glu Gly Phe Asn Cys Tyr Phe Pro Leu Gln Ser Tyr Gly
485 490 495
Phe Gln Pro Thr Asn Gly Val Gly Tyr Gln Pro Tyr Arg Val Val Val
500 505 510
Leu Ser Phe Glu Leu Leu His Ala Pro Ala Thr Val Cys Gly Pro Lys
515 520 525
Lys Ser Thr Asn Leu Val Lys Asn Lys Cys Val Asn Phe Asn Phe Asn
530 535 540
Gly Leu Thr Gly Thr Gly Val Leu Thr Glu Ser Asn Lys Lys Phe Leu
545 550 555 560
Pro Phe Gln Gln Phe Gly Arg Asp Ile Ala Asp Thr Thr Asp Ala Val
565 570 575
Arg Asp Pro Gln Thr Leu Glu Ile Leu Asp Ile Thr Pro Cys Ser Phe
580 585 590
Gly Gly Val Ser Val Ile Thr Pro Gly Thr Asn Thr Ser Asn Gln Val
595 600 605
Ala Val Leu Tyr Gln Asp Val Asn Cys Thr Glu Val Pro Val Ala Ile
610 615 620
His Ala Asp Gln Leu Thr Pro Thr Trp Arg Val Tyr Ser Thr Gly Ser
625 630 635 640
Asn Val Phe Gln Thr Arg Ala Gly Cys Leu Ile Gly Ala Glu His Val
645 650 655
Asn Asn Ser Tyr Glu Cys Asp Ile Pro Ile Gly Ala Gly Ile Cys Ala
660 665 670
Ser Tyr Gln Thr Gln Thr Asn Ser Pro Arg Arg Ala Arg Ser Val Ala
675 680 685
Ser Gln Ser Ile Ile Ala Tyr Thr Met Ser Leu Gly Ala Glu Asn Ser
690 695 700
Val Ala Tyr Ser Asn Asn Ser Ile Ala Ile Pro Thr Asn Phe Thr Ile
705 710 715 720
Ser Val Thr Thr Glu Ile Leu Pro Val Ser Met Thr Lys Thr Ser Val
725 730 735
Asp Cys Thr Met Tyr Ile Cys Gly Asp Ser Thr Glu Cys Ser Asn Leu
740 745 750
Leu Leu Gln Tyr Gly Ser Phe Cys Thr Gln Leu Asn Arg Ala Leu Thr
755 760 765
Gly Ile Ala Val Glu Gln Asp Lys Asn Thr Gln Glu Val Phe Ala Gln
770 775 780
Val Lys Gln Ile Tyr Lys Thr Pro Pro Ile Lys Asp Phe Gly Gly Phe
785 790 795 800
Asn Phe Ser Gln Ile Leu Pro Asp Pro Ser Lys Pro Ser Lys Arg Ser
805 810 815
Phe Ile Glu Asp Leu Leu Phe Asn Lys Val Thr Leu Ala Asp Ala Gly
820 825 830
Phe Ile Lys Gln Tyr Gly Asp Cys Leu Gly Asp Ile Ala Ala Arg Asp
835 840 845
Leu Ile Cys Ala Gln Lys Phe Asn Gly Leu Thr Val Leu Pro Pro Leu
850 855 860
Leu Thr Asp Glu Met Ile Ala Gln Tyr Thr Ser Ala Leu Leu Ala Gly
865 870 875 880
Thr Ile Thr Ser Gly Trp Thr Phe Gly Ala Gly Ala Ala Leu Gln Ile
885 890 895
Pro Phe Ala Met Gln Met Ala Tyr Arg Phe Asn Gly Ile Gly Val Thr
900 905 910
Gln Asn Val Leu Tyr Glu Asn Gln Lys Leu Ile Ala Asn Gln Phe Asn
915 920 925
Ser Ala Ile Gly Lys Ile Gln Asp Ser Leu Ser Ser Thr Ala Ser Ala
930 935 940
Leu Gly Lys Leu Gln Asp Val Val Asn Gln Asn Ala Gln Ala Leu Asn
945 950 955 960
Thr Leu Val Lys Gln Leu Ser Ser Asn Phe Gly Ala Ile Ser Ser Val
965 970 975
Leu Asn Asp Ile Leu Ser Arg Leu Asp Pro Pro Glu Ala Glu Val Gln
980 985 990
Ile Asp Arg Leu Ile Thr Gly Arg Leu Gln Ser Leu Gln Thr Tyr Val
995 1000 1005
Thr Gln Gln Leu Ile Arg Ala Ala Glu Ile Arg Ala Ser Ala Asn
1010 1015 1020
Leu Ala Ala Thr Lys Met Ser Glu Cys Val Leu Gly Gln Ser Lys
1025 1030 1035
Arg Val Asp Phe Cys Gly Lys Gly Tyr His Leu Met Ser Phe Pro
1040 1045 1050
Gln Ser Ala Pro His Gly Val Val Phe Leu His Val Thr Tyr Val
1055 1060 1065
Pro Ala Gln Glu Lys Asn Phe Thr Thr Ala Pro Ala Ile Cys His
1070 1075 1080
Asp Gly Lys Ala His Phe Pro Arg Glu Gly Val Phe Val Ser Asn
1085 1090 1095
Gly Thr His Trp Phe Val Thr Gln Arg Asn Phe Tyr Glu Pro Gln
1100 1105 1110
Ile Ile Thr Thr Asp Asn Thr Phe Val Ser Gly Asn Cys Asp Val
1115 1120 1125
Val Ile Gly Ile Val Asn Asn Thr Val Tyr Asp Pro Leu Gln Pro
1130 1135 1140
Glu Leu Asp Ser Phe Lys Glu Glu Leu Asp Lys Tyr Phe Lys Asn
1145 1150 1155
His Thr Ser Pro Asp Val Asp Leu Gly Asp Ile Ser Gly Ile Asn
1160 1165 1170
Ala Ser Val Val Asn Ile Gln Lys Glu Ile Asp Arg Leu Asn Glu
1175 1180 1185
Val Ala Lys Asn Leu Asn Glu Ser Leu Ile Asp Leu Gln Glu Leu
1190 1195 1200
Gly Lys Tyr Glu Gln Tyr Ile Lys Trp Pro Trp Tyr Ile Trp Leu
1205 1210 1215
Gly Phe Ile Ala Gly Leu Ile Ala Ile Val Met Val Thr Ile Met
1220 1225 1230
Leu Cys Cys Met Thr Ser Cys Cys Ser Cys Leu Lys Gly Cys Cys
1235 1240 1245
Ser Cys Gly Ser Cys Cys Lys Phe Asp Glu Asp Asp Ser Glu Pro
1250 1255 1260
Val Leu Lys Gly Val Lys Leu His Tyr Thr
1265 1270
<210> 5
<211> 3822
<212> DNA
<213> Artificial Sequence
<220>
<223> S-(deg-S2)
<400> 5
atgttcgtct tcctggtcct gctgcctctg gtctcctcac agtgcgtcaa tctgacaact 60
cggactcagc tgccacctgc ttatactaat agcttcacca gaggcgtgta ctatcctgac 120
aaggtgttta gaagctccgt gctgcactct acacaggatc tgtttctgcc attctttagc 180
aacgtgacct ggttccacgc catccacgtg agcggcacca atggcacaaa gcggttcgac 240
aatcccgtgc tgccttttaa cgatggcgtg tacttcgcct ctaccgagaa gagcaacatc 300
atcagaggct ggatctttgg caccacactg gactccaaga cacagtctct gctgatcgtg 360
aacaatgcca ccaacgtggt catcaaggtg tgcgagttcc agttttgtaa tgatcccttc 420
ctgggcgtgt actatcacaa gaacaataag agctggatgg agtccgagtt tagagtgtat 480
tctagcgcca acaactgcac atttgagtac gtgagccagc ctttcctgat ggacctggag 540
ggcaagcagg gcaatttcaa gaacctgagg gagttcgtgt ttaagaatat cgacggctac 600
ttcaaaatct actctaagca cacccccatc aacctggtgc gcgacctgcc tcagggcttc 660
agcgccctgg agcccctggt ggatctgcct atcggcatca acatcacccg gtttcagaca 720
ctgctggccc tgcacagaag ctacctgaca cccggcgact cctctagcgg atggaccgcc 780
ggcgctgccg cctactatgt gggctacctc cagccccgga ccttcctgct gaagtacaac 840
gagaatggca ccatcacaga cgcagtggat tgcgccctgg accccctgag cgagacaaag 900
tgtacactga agtcctttac cgtggagaag ggcatctatc agacatccaa tttcagggtg 960
cagccaaccg agtctatcgt gcgctttcct aatatcacaa acctgtgccc atttggcgag 1020
gtgttcaacg caacccgctt cgccagcgtg tacgcctgga ataggaagcg gatcagcaac 1080
tgcgtggccg actatagcgt gctgtacaac tccgcctctt tcagcacctt taagtgctat 1140
ggcgtgtccc ccacaaagct gaatgacctg tgctttacca acgtctacgc cgattctttc 1200
gtgatcaggg gcgacgaggt gcgccagatc gcccccggcc agacaggcaa gatcgcagac 1260
tacaattata agctgccaga cgatttcacc ggctgcgtga tcgcctggaa cagcaacaat 1320
ctggattcca aagtgggcgg caactacaat tatctgtacc ggctgtttag aaagagcaat 1380
ctgaagccct tcgagaggga catctctaca gaaatctacc aggccggcag caccccttgc 1440
aatggcgtgg agggctttaa ctgttatttc ccactccagt cctacggctt ccagcccaca 1500
aacggcgtgg gctatcagcc ttaccgcgtg gtggtgctga gctttgagct gctgcacgcc 1560
ccagcaacag tgtgcggccc caagaagtcc accaatctgg tgaagaacaa gtgcgtgaac 1620
ttcaacttca acggcctgac cggcacaggc gtgctgaccg agtccaacaa gaagttcctg 1680
ccatttcagc agttcggcag ggacatcgca gataccacag acgccgtgcg cgacccacag 1740
accctggaga tcctggacat cacaccctgc tctttcggcg gcgtgagcgt gatcacaccc 1800
ggcaccaata caagcaacca ggtggccgtg ctgtatcagg acgtgaattg taccgaggtg 1860
cccgtggcta tccacgccga tcagctgacc ccaacatggc gggtgtacag caccggctcc 1920
aacgtcttcc agacaagagc cggatgcctg atcggagcag agcacgtgaa caattcctat 1980
gagtgcgaca tcccaatcgg cgccggcatc tgtgcctctt accagaccca gacaaactct 2040
cccagaagag cccggagcgt ggcctcccag tctatcatcg cctataccat gtccctgggc 2100
gccgagaaca gcgtggccta ctctcagaat agcatcgcca tcccaaccca gttcacaatc 2160
tctgtgacca cagagatcct gcccgtgtcc atgaccaaga catctgtgga ctgcacaatg 2220
tatatctgtg gcgattctac cgagtgcagc aacctgctgc tccagtacgg cagcttttgt 2280
acccagctga atagagccct gacaggcatc gccgtggagc aggataagaa cacacaggag 2340
gtgttcgccc aggtgaagca aatctacaag acccccccta tcaaggactt tggcggcttc 2400
caattttccc agatcctgcc tgatccatcc aagccttcta agcggagctt tatcgaggac 2460
ctgctgttca acaaggtgac cctggccgat gccggcttca tcaagcagta tggcgattgc 2520
ctgggcgaca tcgcagccag ggacctgatc tgcgcccaga agtttaatgg cctgaccgtg 2580
ctgccacccc tgctgacaga tgagatgatc gcacagtaca caagcgccct gctggccggc 2640
accatcacat ccggatggac cttcggcgca ggagccgccc tccagatccc ctttgccatg 2700
cagatggcct ataggttcaa cggcatcggc gtgacccaga atgtgctgta cgagaaccag 2760
aagctgatcg ccaatcagtt taactccgcc atcggcaaga tccaggacag cctgtcctct 2820
acagccagcg ccctgggcaa gctccaggat gtggtgaatc agaacgccca ggccctgaat 2880
accctggtga agcagctgag cagcaacttc ggcgccatct ctagcgtgct gaatgacatc 2940
ctgagccggc tggacaaggt ggaggcagag gtgcagatcg accggctgat caccggccgg 3000
ctccagagcc tccagaccta tgtgacacag cagctgatca gggccgccga gatcagggcc 3060
agcgccaatc tggcagcaac caagatgtcc gagtgcgtgc tgggccagtc taagagagtg 3120
gacttttgtg gcaagggcta tcacctgatg tccttccctc agtctgcccc acacggcgtg 3180
gtgtttctgc acgtgaccta cgtgcccgcc caggagaagc agttcaccac agcccctgcc 3240
atctgccacg atggcaaggc ccactttcca agggagggcg tgttcgtgtc ccagggcacc 3300
cactggtttg tgacacagcg caatttctac gagccccaga tcatcaccac agacaacacc 3360
ttcgtgagcg gcaactgtga cgtggtcatc ggcatcgtgc agaataccgt gtatgatcca 3420
ctccagcccg agctggacag ctttaaggag gagctggata agtatttcaa gcaacacacc 3480
tcccctgacg tggatctggg cgacatcagc ggcatccaag cctccgtggt gaacatccag 3540
aaggagatcg accgcctgaa cgaggtggct aagaatctgc aggagagcct gatcgacctc 3600
caggagctgg gcaagtatga gcagtacatc aagtggccct ggtacatctg gctgggcttc 3660
atcgccggcc tgatcgccat cgtgatggtg accatcatgc tgtgctgtat gacatcctgc 3720
tgttcttgcc tgaagggctg ctgtagctgt ggctcctgct gtaagtttga cgaggatgac 3780
tctgaacctg tgctgaaggg cgtgaagctg cattacacct aa 3822
<210> 6
<211> 1273
<212> PRT
<213> Artificial Sequence
<220>
<223> S-(deg-S2)
<400> 6
Met Phe Val Phe Leu Val Leu Leu Pro Leu Val Ser Ser Gln Cys Val
1 5 10 15
Asn Leu Thr Thr Arg Thr Gln Leu Pro Pro Ala Tyr Thr Asn Ser Phe
20 25 30
Thr Arg Gly Val Tyr Tyr Pro Asp Lys Val Phe Arg Ser Ser Val Leu
35 40 45
His Ser Thr Gln Asp Leu Phe Leu Pro Phe Phe Ser Asn Val Thr Trp
50 55 60
Phe His Ala Ile His Val Ser Gly Thr Asn Gly Thr Lys Arg Phe Asp
65 70 75 80
Asn Pro Val Leu Pro Phe Asn Asp Gly Val Tyr Phe Ala Ser Thr Glu
85 90 95
Lys Ser Asn Ile Ile Arg Gly Trp Ile Phe Gly Thr Thr Leu Asp Ser
100 105 110
Lys Thr Gln Ser Leu Leu Ile Val Asn Asn Ala Thr Asn Val Val Ile
115 120 125
Lys Val Cys Glu Phe Gln Phe Cys Asn Asp Pro Phe Leu Gly Val Tyr
130 135 140
Tyr His Lys Asn Asn Lys Ser Trp Met Glu Ser Glu Phe Arg Val Tyr
145 150 155 160
Ser Ser Ala Asn Asn Cys Thr Phe Glu Tyr Val Ser Gln Pro Phe Leu
165 170 175
Met Asp Leu Glu Gly Lys Gln Gly Asn Phe Lys Asn Leu Arg Glu Phe
180 185 190
Val Phe Lys Asn Ile Asp Gly Tyr Phe Lys Ile Tyr Ser Lys His Thr
195 200 205
Pro Ile Asn Leu Val Arg Asp Leu Pro Gln Gly Phe Ser Ala Leu Glu
210 215 220
Pro Leu Val Asp Leu Pro Ile Gly Ile Asn Ile Thr Arg Phe Gln Thr
225 230 235 240
Leu Leu Ala Leu His Arg Ser Tyr Leu Thr Pro Gly Asp Ser Ser Ser
245 250 255
Gly Trp Thr Ala Gly Ala Ala Ala Tyr Tyr Val Gly Tyr Leu Gln Pro
260 265 270
Arg Thr Phe Leu Leu Lys Tyr Asn Glu Asn Gly Thr Ile Thr Asp Ala
275 280 285
Val Asp Cys Ala Leu Asp Pro Leu Ser Glu Thr Lys Cys Thr Leu Lys
290 295 300
Ser Phe Thr Val Glu Lys Gly Ile Tyr Gln Thr Ser Asn Phe Arg Val
305 310 315 320
Gln Pro Thr Glu Ser Ile Val Arg Phe Pro Asn Ile Thr Asn Leu Cys
325 330 335
Pro Phe Gly Glu Val Phe Asn Ala Thr Arg Phe Ala Ser Val Tyr Ala
340 345 350
Trp Asn Arg Lys Arg Ile Ser Asn Cys Val Ala Asp Tyr Ser Val Leu
355 360 365
Tyr Asn Ser Ala Ser Phe Ser Thr Phe Lys Cys Tyr Gly Val Ser Pro
370 375 380
Thr Lys Leu Asn Asp Leu Cys Phe Thr Asn Val Tyr Ala Asp Ser Phe
385 390 395 400
Val Ile Arg Gly Asp Glu Val Arg Gln Ile Ala Pro Gly Gln Thr Gly
405 410 415
Lys Ile Ala Asp Tyr Asn Tyr Lys Leu Pro Asp Asp Phe Thr Gly Cys
420 425 430
Val Ile Ala Trp Asn Ser Asn Asn Leu Asp Ser Lys Val Gly Gly Asn
435 440 445
Tyr Asn Tyr Leu Tyr Arg Leu Phe Arg Lys Ser Asn Leu Lys Pro Phe
450 455 460
Glu Arg Asp Ile Ser Thr Glu Ile Tyr Gln Ala Gly Ser Thr Pro Cys
465 470 475 480
Asn Gly Val Glu Gly Phe Asn Cys Tyr Phe Pro Leu Gln Ser Tyr Gly
485 490 495
Phe Gln Pro Thr Asn Gly Val Gly Tyr Gln Pro Tyr Arg Val Val Val
500 505 510
Leu Ser Phe Glu Leu Leu His Ala Pro Ala Thr Val Cys Gly Pro Lys
515 520 525
Lys Ser Thr Asn Leu Val Lys Asn Lys Cys Val Asn Phe Asn Phe Asn
530 535 540
Gly Leu Thr Gly Thr Gly Val Leu Thr Glu Ser Asn Lys Lys Phe Leu
545 550 555 560
Pro Phe Gln Gln Phe Gly Arg Asp Ile Ala Asp Thr Thr Asp Ala Val
565 570 575
Arg Asp Pro Gln Thr Leu Glu Ile Leu Asp Ile Thr Pro Cys Ser Phe
580 585 590
Gly Gly Val Ser Val Ile Thr Pro Gly Thr Asn Thr Ser Asn Gln Val
595 600 605
Ala Val Leu Tyr Gln Asp Val Asn Cys Thr Glu Val Pro Val Ala Ile
610 615 620
His Ala Asp Gln Leu Thr Pro Thr Trp Arg Val Tyr Ser Thr Gly Ser
625 630 635 640
Asn Val Phe Gln Thr Arg Ala Gly Cys Leu Ile Gly Ala Glu His Val
645 650 655
Asn Asn Ser Tyr Glu Cys Asp Ile Pro Ile Gly Ala Gly Ile Cys Ala
660 665 670
Ser Tyr Gln Thr Gln Thr Asn Ser Pro Arg Arg Ala Arg Ser Val Ala
675 680 685
Ser Gln Ser Ile Ile Ala Tyr Thr Met Ser Leu Gly Ala Glu Asn Ser
690 695 700
Val Ala Tyr Ser Gln Asn Ser Ile Ala Ile Pro Thr Gln Phe Thr Ile
705 710 715 720
Ser Val Thr Thr Glu Ile Leu Pro Val Ser Met Thr Lys Thr Ser Val
725 730 735
Asp Cys Thr Met Tyr Ile Cys Gly Asp Ser Thr Glu Cys Ser Asn Leu
740 745 750
Leu Leu Gln Tyr Gly Ser Phe Cys Thr Gln Leu Asn Arg Ala Leu Thr
755 760 765
Gly Ile Ala Val Glu Gln Asp Lys Asn Thr Gln Glu Val Phe Ala Gln
770 775 780
Val Lys Gln Ile Tyr Lys Thr Pro Pro Ile Lys Asp Phe Gly Gly Phe
785 790 795 800
Gln Phe Ser Gln Ile Leu Pro Asp Pro Ser Lys Pro Ser Lys Arg Ser
805 810 815
Phe Ile Glu Asp Leu Leu Phe Asn Lys Val Thr Leu Ala Asp Ala Gly
820 825 830
Phe Ile Lys Gln Tyr Gly Asp Cys Leu Gly Asp Ile Ala Ala Arg Asp
835 840 845
Leu Ile Cys Ala Gln Lys Phe Asn Gly Leu Thr Val Leu Pro Pro Leu
850 855 860
Leu Thr Asp Glu Met Ile Ala Gln Tyr Thr Ser Ala Leu Leu Ala Gly
865 870 875 880
Thr Ile Thr Ser Gly Trp Thr Phe Gly Ala Gly Ala Ala Leu Gln Ile
885 890 895
Pro Phe Ala Met Gln Met Ala Tyr Arg Phe Asn Gly Ile Gly Val Thr
900 905 910
Gln Asn Val Leu Tyr Glu Asn Gln Lys Leu Ile Ala Asn Gln Phe Asn
915 920 925
Ser Ala Ile Gly Lys Ile Gln Asp Ser Leu Ser Ser Thr Ala Ser Ala
930 935 940
Leu Gly Lys Leu Gln Asp Val Val Asn Gln Asn Ala Gln Ala Leu Asn
945 950 955 960
Thr Leu Val Lys Gln Leu Ser Ser Asn Phe Gly Ala Ile Ser Ser Val
965 970 975
Leu Asn Asp Ile Leu Ser Arg Leu Asp Pro Pro Glu Ala Glu Val Gln
980 985 990
Ile Asp Arg Leu Ile Thr Gly Arg Leu Gln Ser Leu Gln Thr Tyr Val
995 1000 1005
Thr Gln Gln Leu Ile Arg Ala Ala Glu Ile Arg Ala Ser Ala Asn
1010 1015 1020
Leu Ala Ala Thr Lys Met Ser Glu Cys Val Leu Gly Gln Ser Lys
1025 1030 1035
Arg Val Asp Phe Cys Gly Lys Gly Tyr His Leu Met Ser Phe Pro
1040 1045 1050
Gln Ser Ala Pro His Gly Val Val Phe Leu His Val Thr Tyr Val
1055 1060 1065
Pro Ala Gln Glu Lys Gln Phe Thr Thr Ala Pro Ala Ile Cys His
1070 1075 1080
Asp Gly Lys Ala His Phe Pro Arg Glu Gly Val Phe Val Ser Gln
1085 1090 1095
Gly Thr His Trp Phe Val Thr Gln Arg Asn Phe Tyr Glu Pro Gln
1100 1105 1110
Ile Ile Thr Thr Asp Asn Thr Phe Val Ser Gly Asn Cys Asp Val
1115 1120 1125
Val Ile Gly Ile Val Gln Asn Thr Val Tyr Asp Pro Leu Gln Pro
1130 1135 1140
Glu Leu Asp Ser Phe Lys Glu Glu Leu Asp Lys Tyr Phe Lys Gln
1145 1150 1155
His Thr Ser Pro Asp Val Asp Leu Gly Asp Ile Ser Gly Ile Gln
1160 1165 1170
Ala Ser Val Val Asn Ile Gln Lys Glu Ile Asp Arg Leu Asn Glu
1175 1180 1185
Val Ala Lys Asn Leu Gln Glu Ser Leu Ile Asp Leu Gln Glu Leu
1190 1195 1200
Gly Lys Tyr Glu Gln Tyr Ile Lys Trp Pro Trp Tyr Ile Trp Leu
1205 1210 1215
Gly Phe Ile Ala Gly Leu Ile Ala Ile Val Met Val Thr Ile Met
1220 1225 1230
Leu Cys Cys Met Thr Ser Cys Cys Ser Cys Leu Lys Gly Cys Cys
1235 1240 1245
Ser Cys Gly Ser Cys Cys Lys Phe Asp Glu Asp Asp Ser Glu Pro
1250 1255 1260
Val Leu Lys Gly Val Lys Leu His Tyr Thr
1265 1270
<210> 7
<211> 3822
<212> DNA
<213> Artificial Sequence
<220>
<223> S-(S2-1194)
<400> 7
atgttcgtct tcctggtcct gctgcctctg gtctcctcac agtgcgtcaa tctgacaact 60
cggactcagc tgccacctgc ttatactaat agcttcacca gaggcgtgta ctatcctgac 120
aaggtgttta gaagctccgt gctgcactct acacaggatc tgtttctgcc attctttagc 180
aacgtgacct ggttccacgc catccacgtg agcggcacca atggcacaaa gcggttcgac 240
aatcccgtgc tgccttttaa cgatggcgtg tacttcgcct ctaccgagaa gagcaacatc 300
atcagaggct ggatctttgg caccacactg gactccaaga cacagtctct gctgatcgtg 360
aacaatgcca ccaacgtggt catcaaggtg tgcgagttcc agttttgtaa tgatcccttc 420
ctgggcgtgt actatcacaa gaacaataag agctggatgg agtccgagtt tagagtgtat 480
tctagcgcca acaactgcac atttgagtac gtgagccagc ctttcctgat ggacctggag 540
ggcaagcagg gcaatttcaa gaacctgagg gagttcgtgt ttaagaatat cgacggctac 600
ttcaaaatct actctaagca cacccccatc aacctggtgc gcgacctgcc tcagggcttc 660
agcgccctgg agcccctggt ggatctgcct atcggcatca acatcacccg gtttcagaca 720
ctgctggccc tgcacagaag ctacctgaca cccggcgact cctctagcgg atggaccgcc 780
ggcgctgccg cctactatgt gggctacctc cagccccgga ccttcctgct gaagtacaac 840
gagaatggca ccatcacaga cgcagtggat tgcgccctgg accccctgag cgagacaaag 900
tgtacactga agtcctttac cgtggagaag ggcatctatc agacatccaa tttcagggtg 960
cagccaaccg agtctatcgt gcgctttcct aatatcacaa acctgtgccc atttggcgag 1020
gtgttcaacg caacccgctt cgccagcgtg tacgcctgga ataggaagcg gatcagcaac 1080
tgcgtggccg actatagcgt gctgtacaac tccgcctctt tcagcacctt taagtgctat 1140
ggcgtgtccc ccacaaagct gaatgacctg tgctttacca acgtctacgc cgattctttc 1200
gtgatcaggg gcgacgaggt gcgccagatc gcccccggcc agacaggcaa gatcgcagac 1260
tacaattata agctgccaga cgatttcacc ggctgcgtga tcgcctggaa cagcaacaat 1320
ctggattcca aagtgggcgg caactacaat tatctgtacc ggctgtttag aaagagcaat 1380
ctgaagccct tcgagaggga catctctaca gaaatctacc aggccggcag caccccttgc 1440
aatggcgtgg agggctttaa ctgttatttc ccactccagt cctacggctt ccagcccaca 1500
aacggcgtgg gctatcagcc ttaccgcgtg gtggtgctga gctttgagct gctgcacgcc 1560
ccagcaacag tgtgcggccc caagaagtcc accaatctgg tgaagaacaa gtgcgtgaac 1620
ttcaacttca acggcctgac cggcacaggc gtgctgaccg agtccaacaa gaagttcctg 1680
ccatttcagc agttcggcag ggacatcgca gataccacag acgccgtgcg cgacccacag 1740
accctggaga tcctggacat cacaccctgc tctttcggcg gcgtgagcgt gatcacaccc 1800
ggcaccaata caagcaacca ggtggccgtg ctgtatcagg acgtgaattg taccgaggtg 1860
cccgtggcta tccacgccga tcagctgacc ccaacatggc gggtgtacag caccggctcc 1920
aacgtcttcc agacaagagc cggatgcctg atcggagcag agcacgtgaa caattcctat 1980
gagtgcgaca tcccaatcgg cgccggcatc tgtgcctctt accagaccca gacaaactct 2040
cccagaagag cccggagcgt ggcctcccag tctatcatcg cctataccat gtccctgggc 2100
gccgagaaca gcgtggccta ctctcagaat agcatcgcca tcccaaccca gttcacaatc 2160
tctgtgacca cagagatcct gcccgtgtcc atgaccaaga catctgtgga ctgcacaatg 2220
tatatctgtg gcgattctac cgagtgcagc aacctgctgc tccagtacgg cagcttttgt 2280
acccagctga atagagccct gacaggcatc gccgtggagc aggataagaa cacacaggag 2340
gtgttcgccc aggtgaagca aatctacaag acccccccta tcaaggactt tggcggcttc 2400
caattttccc agatcctgcc tgatccatcc aagccttcta agcggagctt tatcgaggac 2460
ctgctgttca acaaggtgac cctggccgat gccggcttca tcaagcagta tggcgattgc 2520
ctgggcgaca tcgcagccag ggacctgatc tgcgcccaga agtttaatgg cctgaccgtg 2580
ctgccacccc tgctgacaga tgagatgatc gcacagtaca caagcgccct gctggccggc 2640
accatcacat ccggatggac cttcggcgca ggagccgccc tccagatccc ctttgccatg 2700
cagatggcct ataggttcaa cggcatcggc gtgacccaga atgtgctgta cgagaaccag 2760
aagctgatcg ccaatcagtt taactccgcc atcggcaaga tccaggacag cctgtcctct 2820
acagccagcg ccctgggcaa gctccaggat gtggtgaatc agaacgccca ggccctgaat 2880
accctggtga agcagctgag cagcaacttc ggcgccatct ctagcgtgct gaatgacatc 2940
ctgagccggc tggacaaggt ggaggcagag gtgcagatcg accggctgat caccggccgg 3000
ctccagagcc tccagaccta tgtgacacag cagctgatca gggccgccga gatcagggcc 3060
agcgccaatc tggcagcaac caagatgtcc gagtgcgtgc tgggccagtc taagagagtg 3120
gacttttgtg gcaagggcta tcacctgatg tccttccctc agtctgcccc acacggcgtg 3180
gtgtttctgc acgtgaccta cgtgcccgcc caggagaagc agttcaccac agcccctgcc 3240
atctgccacg atggcaaggc ccactttcca agggagggcg tgttcgtgtc ccagggcacc 3300
cactggtttg tgacacagcg caatttctac gagccccaga tcatcaccac agacaacacc 3360
ttcgtgagcg gcaactgtga cgtggtcatc ggcatcgtgc agaataccgt gtatgatcca 3420
ctccagcccg agctggacag ctttaaggag gagctggata agtatttcaa gcaacacacc 3480
tcccctgacg tggatctggg cgacatcagc ggcatccaag cctccgtggt gaacatccag 3540
aaggagatcg accgcctgaa cgaggtggct aagaatctga acgagagcct gatcgacctc 3600
caggagctgg gcaagtatga gcagtacatc aagtggccct ggtacatctg gctgggcttc 3660
atcgccggcc tgatcgccat cgtgatggtg accatcatgc tgtgctgtat gacatcctgc 3720
tgttcttgcc tgaagggctg ctgtagctgt ggctcctgct gtaagtttga cgaggatgac 3780
tctgaacctg tgctgaaggg cgtgaagctg cattacacct aa 3822
<210> 8
<211> 1273
<212> PRT
<213> Artificial Sequence
<220>
<223> S-(S2-1194)
<400> 8
Met Phe Val Phe Leu Val Leu Leu Pro Leu Val Ser Ser Gln Cys Val
1 5 10 15
Asn Leu Thr Thr Arg Thr Gln Leu Pro Pro Ala Tyr Thr Asn Ser Phe
20 25 30
Thr Arg Gly Val Tyr Tyr Pro Asp Lys Val Phe Arg Ser Ser Val Leu
35 40 45
His Ser Thr Gln Asp Leu Phe Leu Pro Phe Phe Ser Asn Val Thr Trp
50 55 60
Phe His Ala Ile His Val Ser Gly Thr Asn Gly Thr Lys Arg Phe Asp
65 70 75 80
Asn Pro Val Leu Pro Phe Asn Asp Gly Val Tyr Phe Ala Ser Thr Glu
85 90 95
Lys Ser Asn Ile Ile Arg Gly Trp Ile Phe Gly Thr Thr Leu Asp Ser
100 105 110
Lys Thr Gln Ser Leu Leu Ile Val Asn Asn Ala Thr Asn Val Val Ile
115 120 125
Lys Val Cys Glu Phe Gln Phe Cys Asn Asp Pro Phe Leu Gly Val Tyr
130 135 140
Tyr His Lys Asn Asn Lys Ser Trp Met Glu Ser Glu Phe Arg Val Tyr
145 150 155 160
Ser Ser Ala Asn Asn Cys Thr Phe Glu Tyr Val Ser Gln Pro Phe Leu
165 170 175
Met Asp Leu Glu Gly Lys Gln Gly Asn Phe Lys Asn Leu Arg Glu Phe
180 185 190
Val Phe Lys Asn Ile Asp Gly Tyr Phe Lys Ile Tyr Ser Lys His Thr
195 200 205
Pro Ile Asn Leu Val Arg Asp Leu Pro Gln Gly Phe Ser Ala Leu Glu
210 215 220
Pro Leu Val Asp Leu Pro Ile Gly Ile Asn Ile Thr Arg Phe Gln Thr
225 230 235 240
Leu Leu Ala Leu His Arg Ser Tyr Leu Thr Pro Gly Asp Ser Ser Ser
245 250 255
Gly Trp Thr Ala Gly Ala Ala Ala Tyr Tyr Val Gly Tyr Leu Gln Pro
260 265 270
Arg Thr Phe Leu Leu Lys Tyr Asn Glu Asn Gly Thr Ile Thr Asp Ala
275 280 285
Val Asp Cys Ala Leu Asp Pro Leu Ser Glu Thr Lys Cys Thr Leu Lys
290 295 300
Ser Phe Thr Val Glu Lys Gly Ile Tyr Gln Thr Ser Asn Phe Arg Val
305 310 315 320
Gln Pro Thr Glu Ser Ile Val Arg Phe Pro Asn Ile Thr Asn Leu Cys
325 330 335
Pro Phe Gly Glu Val Phe Asn Ala Thr Arg Phe Ala Ser Val Tyr Ala
340 345 350
Trp Asn Arg Lys Arg Ile Ser Asn Cys Val Ala Asp Tyr Ser Val Leu
355 360 365
Tyr Asn Ser Ala Ser Phe Ser Thr Phe Lys Cys Tyr Gly Val Ser Pro
370 375 380
Thr Lys Leu Asn Asp Leu Cys Phe Thr Asn Val Tyr Ala Asp Ser Phe
385 390 395 400
Val Ile Arg Gly Asp Glu Val Arg Gln Ile Ala Pro Gly Gln Thr Gly
405 410 415
Lys Ile Ala Asp Tyr Asn Tyr Lys Leu Pro Asp Asp Phe Thr Gly Cys
420 425 430
Val Ile Ala Trp Asn Ser Asn Asn Leu Asp Ser Lys Val Gly Gly Asn
435 440 445
Tyr Asn Tyr Leu Tyr Arg Leu Phe Arg Lys Ser Asn Leu Lys Pro Phe
450 455 460
Glu Arg Asp Ile Ser Thr Glu Ile Tyr Gln Ala Gly Ser Thr Pro Cys
465 470 475 480
Asn Gly Val Glu Gly Phe Asn Cys Tyr Phe Pro Leu Gln Ser Tyr Gly
485 490 495
Phe Gln Pro Thr Asn Gly Val Gly Tyr Gln Pro Tyr Arg Val Val Val
500 505 510
Leu Ser Phe Glu Leu Leu His Ala Pro Ala Thr Val Cys Gly Pro Lys
515 520 525
Lys Ser Thr Asn Leu Val Lys Asn Lys Cys Val Asn Phe Asn Phe Asn
530 535 540
Gly Leu Thr Gly Thr Gly Val Leu Thr Glu Ser Asn Lys Lys Phe Leu
545 550 555 560
Pro Phe Gln Gln Phe Gly Arg Asp Ile Ala Asp Thr Thr Asp Ala Val
565 570 575
Arg Asp Pro Gln Thr Leu Glu Ile Leu Asp Ile Thr Pro Cys Ser Phe
580 585 590
Gly Gly Val Ser Val Ile Thr Pro Gly Thr Asn Thr Ser Asn Gln Val
595 600 605
Ala Val Leu Tyr Gln Asp Val Asn Cys Thr Glu Val Pro Val Ala Ile
610 615 620
His Ala Asp Gln Leu Thr Pro Thr Trp Arg Val Tyr Ser Thr Gly Ser
625 630 635 640
Asn Val Phe Gln Thr Arg Ala Gly Cys Leu Ile Gly Ala Glu His Val
645 650 655
Asn Asn Ser Tyr Glu Cys Asp Ile Pro Ile Gly Ala Gly Ile Cys Ala
660 665 670
Ser Tyr Gln Thr Gln Thr Asn Ser Pro Arg Arg Ala Arg Ser Val Ala
675 680 685
Ser Gln Ser Ile Ile Ala Tyr Thr Met Ser Leu Gly Ala Glu Asn Ser
690 695 700
Val Ala Tyr Ser Gln Asn Ser Ile Ala Ile Pro Thr Gln Phe Thr Ile
705 710 715 720
Ser Val Thr Thr Glu Ile Leu Pro Val Ser Met Thr Lys Thr Ser Val
725 730 735
Asp Cys Thr Met Tyr Ile Cys Gly Asp Ser Thr Glu Cys Ser Asn Leu
740 745 750
Leu Leu Gln Tyr Gly Ser Phe Cys Thr Gln Leu Asn Arg Ala Leu Thr
755 760 765
Gly Ile Ala Val Glu Gln Asp Lys Asn Thr Gln Glu Val Phe Ala Gln
770 775 780
Val Lys Gln Ile Tyr Lys Thr Pro Pro Ile Lys Asp Phe Gly Gly Phe
785 790 795 800
Gln Phe Ser Gln Ile Leu Pro Asp Pro Ser Lys Pro Ser Lys Arg Ser
805 810 815
Phe Ile Glu Asp Leu Leu Phe Asn Lys Val Thr Leu Ala Asp Ala Gly
820 825 830
Phe Ile Lys Gln Tyr Gly Asp Cys Leu Gly Asp Ile Ala Ala Arg Asp
835 840 845
Leu Ile Cys Ala Gln Lys Phe Asn Gly Leu Thr Val Leu Pro Pro Leu
850 855 860
Leu Thr Asp Glu Met Ile Ala Gln Tyr Thr Ser Ala Leu Leu Ala Gly
865 870 875 880
Thr Ile Thr Ser Gly Trp Thr Phe Gly Ala Gly Ala Ala Leu Gln Ile
885 890 895
Pro Phe Ala Met Gln Met Ala Tyr Arg Phe Asn Gly Ile Gly Val Thr
900 905 910
Gln Asn Val Leu Tyr Glu Asn Gln Lys Leu Ile Ala Asn Gln Phe Asn
915 920 925
Ser Ala Ile Gly Lys Ile Gln Asp Ser Leu Ser Ser Thr Ala Ser Ala
930 935 940
Leu Gly Lys Leu Gln Asp Val Val Asn Gln Asn Ala Gln Ala Leu Asn
945 950 955 960
Thr Leu Val Lys Gln Leu Ser Ser Asn Phe Gly Ala Ile Ser Ser Val
965 970 975
Leu Asn Asp Ile Leu Ser Arg Leu Asp Pro Pro Glu Ala Glu Val Gln
980 985 990
Ile Asp Arg Leu Ile Thr Gly Arg Leu Gln Ser Leu Gln Thr Tyr Val
995 1000 1005
Thr Gln Gln Leu Ile Arg Ala Ala Glu Ile Arg Ala Ser Ala Asn
1010 1015 1020
Leu Ala Ala Thr Lys Met Ser Glu Cys Val Leu Gly Gln Ser Lys
1025 1030 1035
Arg Val Asp Phe Cys Gly Lys Gly Tyr His Leu Met Ser Phe Pro
1040 1045 1050
Gln Ser Ala Pro His Gly Val Val Phe Leu His Val Thr Tyr Val
1055 1060 1065
Pro Ala Gln Glu Lys Gln Phe Thr Thr Ala Pro Ala Ile Cys His
1070 1075 1080
Asp Gly Lys Ala His Phe Pro Arg Glu Gly Val Phe Val Ser Gln
1085 1090 1095
Gly Thr His Trp Phe Val Thr Gln Arg Asn Phe Tyr Glu Pro Gln
1100 1105 1110
Ile Ile Thr Thr Asp Asn Thr Phe Val Ser Gly Asn Cys Asp Val
1115 1120 1125
Val Ile Gly Ile Val Gln Asn Thr Val Tyr Asp Pro Leu Gln Pro
1130 1135 1140
Glu Leu Asp Ser Phe Lys Glu Glu Leu Asp Lys Tyr Phe Lys Gln
1145 1150 1155
His Thr Ser Pro Asp Val Asp Leu Gly Asp Ile Ser Gly Ile Gln
1160 1165 1170
Ala Ser Val Val Asn Ile Gln Lys Glu Ile Asp Arg Leu Asn Glu
1175 1180 1185
Val Ala Lys Asn Leu Asn Glu Ser Leu Ile Asp Leu Gln Glu Leu
1190 1195 1200
Gly Lys Tyr Glu Gln Tyr Ile Lys Trp Pro Trp Tyr Ile Trp Leu
1205 1210 1215
Gly Phe Ile Ala Gly Leu Ile Ala Ile Val Met Val Thr Ile Met
1220 1225 1230
Leu Cys Cys Met Thr Ser Cys Cys Ser Cys Leu Lys Gly Cys Cys
1235 1240 1245
Ser Cys Gly Ser Cys Cys Lys Phe Asp Glu Asp Asp Ser Glu Pro
1250 1255 1260
Val Leu Lys Gly Val Lys Leu His Tyr Thr
1265 1270
<210> 9
<211> 3822
<212> DNA
<213> Artificial Sequence
<220>
<223> S-(deg-RBD-801)
<400> 9
atgttcgtct tcctggtcct gctgcctctg gtctcctcac agtgcgtcaa tctgacaact 60
cggactcagc tgccacctgc ttatactaat agcttcacca gaggcgtgta ctatcctgac 120
aaggtgttta gaagctccgt gctgcactct acacaggatc tgtttctgcc attctttagc 180
aacgtgacct ggttccacgc catccacgtg agcggcacca atggcacaaa gcggttcgac 240
aatcccgtgc tgccttttaa cgatggcgtg tacttcgcct ctaccgagaa gagcaacatc 300
atcagaggct ggatctttgg caccacactg gactccaaga cacagtctct gctgatcgtg 360
aacaatgcca ccaacgtggt catcaaggtg tgcgagttcc agttttgtaa tgatcccttc 420
ctgggcgtgt actatcacaa gaacaataag agctggatgg agtccgagtt tagagtgtat 480
tctagcgcca acaactgcac atttgagtac gtgagccagc ctttcctgat ggacctggag 540
ggcaagcagg gcaatttcaa gaacctgagg gagttcgtgt ttaagaatat cgacggctac 600
ttcaaaatct actctaagca cacccccatc aacctggtgc gcgacctgcc tcagggcttc 660
agcgccctgg agcccctggt ggatctgcct atcggcatca acatcacccg gtttcagaca 720
ctgctggccc tgcacagaag ctacctgaca cccggcgact cctctagcgg atggaccgcc 780
ggcgctgccg cctactatgt gggctacctc cagccccgga ccttcctgct gaagtacaac 840
gagaatggca ccatcacaga cgcagtggat tgcgccctgg accccctgag cgagacaaag 900
tgtacactga agtcctttac cgtggagaag ggcatctatc agacatccaa tttcagggtg 960
cagccagccg aggcgatcgt gcgctttcct gaaatcacaa acctgtgccc atttggcgag 1020
gtgttcgagg caacccgctt cgccagcgtg tacgcctgga ataggaagcg gatcagcaac 1080
tgcgtggccg actatagcgt gctgtacaac tccgcctctt tcagcacctt taagtgctat 1140
ggcgtgtccc ccacaaagct gaatgacctg tgctttacca acgtctacgc cgattctttc 1200
gtgatcaggg gcgacgaggt gcgccagatc gcccccggcc agacaggcaa gatcgcagac 1260
tacaattata agctgccaga cgatttcacc ggctgcgtga tcgcctggaa cagcaacaat 1320
ctggattcca aagtgggcgg caactacaat tatctgtacc ggctgtttag aaagagcaat 1380
ctgaagccct tcgagaggga catctctaca gaaatctacc aggccggcag caccccttgc 1440
aatggcgtgg agggctttaa ctgttatttc ccactccagt cctacggctt ccagcccaca 1500
aacggcgtgg gctatcagcc ttaccgcgtg gtggtgctga gctttgagct gctgcacgcc 1560
ccagcaacag tgtgcggccc caagaagtcc accaatctgg tgaagaacaa gtgcgtgaac 1620
ttcaacttca acggcctgac cggcacaggc gtgctgaccg agtccaacaa gaagttcctg 1680
ccatttcagc agttcggcag ggacatcgca gataccacag acgccgtgcg cgacccacag 1740
accctggaga tcctggacat cacaccctgc tctttcggcg gcgtgagcgt gatcacaccc 1800
ggcaccaata caagcaacca ggtggccgtg ctgtatcagg acgtgaattg taccgaggtg 1860
cccgtggcta tccacgccga tcagctgacc ccaacatggc gggtgtacag caccggctcc 1920
aacgtcttcc agacaagagc cggatgcctg atcggagcag agcacgtgaa caattcctat 1980
gagtgcgaca tcccaatcgg cgccggcatc tgtgcctctt accagaccca gacaaactct 2040
cccagaagag cccggagcgt ggcctcccag tctatcatcg cctataccat gtccctgggc 2100
gccgagaaca gcgtggccta ctctaacaat agcatcgcca tcccaaccaa cttcacaatc 2160
tctgtgacca cagagatcct gcccgtgtcc atgaccaaga catctgtgga ctgcacaatg 2220
tatatctgtg gcgattctac cgagtgcagc aacctgctgc tccagtacgg cagcttttgt 2280
acccagctga atagagccct gacaggcatc gccgtggagc aggataagaa cacacaggag 2340
gtgttcgccc aggtgaagca aatctacaag acccccccta tcaaggactt tggcggcttc 2400
caattttccc agatcctgcc tgatccatcc aagccttcta agcggagctt tatcgaggac 2460
ctgctgttca acaaggtgac cctggccgat gccggcttca tcaagcagta tggcgattgc 2520
ctgggcgaca tcgcagccag ggacctgatc tgcgcccaga agtttaatgg cctgaccgtg 2580
ctgccacccc tgctgacaga tgagatgatc gcacagtaca caagcgccct gctggccggc 2640
accatcacat ccggatggac cttcggcgca ggagccgccc tccagatccc ctttgccatg 2700
cagatggcct ataggttcaa cggcatcggc gtgacccaga atgtgctgta cgagaaccag 2760
aagctgatcg ccaatcagtt taactccgcc atcggcaaga tccaggacag cctgtcctct 2820
acagccagcg ccctgggcaa gctccaggat gtggtgaatc agaacgccca ggccctgaat 2880
accctggtga agcagctgag cagcaacttc ggcgccatct ctagcgtgct gaatgacatc 2940
ctgagccggc tggacaaggt ggaggcagag gtgcagatcg accggctgat caccggccgg 3000
ctccagagcc tccagaccta tgtgacacag cagctgatca gggccgccga gatcagggcc 3060
agcgccaatc tggcagcaac caagatgtcc gagtgcgtgc tgggccagtc taagagagtg 3120
gacttttgtg gcaagggcta tcacctgatg tccttccctc agtctgcccc acacggcgtg 3180
gtgtttctgc acgtgaccta cgtgcccgcc caggagaaga acttcaccac agcccctgcc 3240
atctgccacg atggcaaggc ccactttcca agggagggcg tgttcgtgtc caacggcacc 3300
cactggtttg tgacacagcg caatttctac gagccccaga tcatcaccac agacaacacc 3360
ttcgtgagcg gcaactgtga cgtggtcatc ggcatcgtga acaataccgt gtatgatcca 3420
ctccagcccg agctggacag ctttaaggag gagctggata agtatttcaa gaatcacacc 3480
tcccctgacg tggatctggg cgacatcagc ggcatcaatg cctccgtggt gaacatccag 3540
aaggagatcg accgcctgaa cgaggtggct aagaatctga acgagagcct gatcgacctc 3600
caggagctgg gcaagtatga gcagtacatc aagtggccct ggtacatctg gctgggcttc 3660
atcgccggcc tgatcgccat cgtgatggtg accatcatgc tgtgctgtat gacatcctgc 3720
tgttcttgcc tgaagggctg ctgtagctgt ggctcctgct gtaagtttga cgaggatgac 3780
tctgaacctg tgctgaaggg cgtgaagctg cattacacct aa 3822
<210> 10
<211> 1273
<212> PRT
<213> Artificial Sequence
<220>
<223> S-(deg-RBD-801)
<400> 10
Met Phe Val Phe Leu Val Leu Leu Pro Leu Val Ser Ser Gln Cys Val
1 5 10 15
Asn Leu Thr Thr Arg Thr Gln Leu Pro Pro Ala Tyr Thr Asn Ser Phe
20 25 30
Thr Arg Gly Val Tyr Tyr Pro Asp Lys Val Phe Arg Ser Ser Val Leu
35 40 45
His Ser Thr Gln Asp Leu Phe Leu Pro Phe Phe Ser Asn Val Thr Trp
50 55 60
Phe His Ala Ile His Val Ser Gly Thr Asn Gly Thr Lys Arg Phe Asp
65 70 75 80
Asn Pro Val Leu Pro Phe Asn Asp Gly Val Tyr Phe Ala Ser Thr Glu
85 90 95
Lys Ser Asn Ile Ile Arg Gly Trp Ile Phe Gly Thr Thr Leu Asp Ser
100 105 110
Lys Thr Gln Ser Leu Leu Ile Val Asn Asn Ala Thr Asn Val Val Ile
115 120 125
Lys Val Cys Glu Phe Gln Phe Cys Asn Asp Pro Phe Leu Gly Val Tyr
130 135 140
Tyr His Lys Asn Asn Lys Ser Trp Met Glu Ser Glu Phe Arg Val Tyr
145 150 155 160
Ser Ser Ala Asn Asn Cys Thr Phe Glu Tyr Val Ser Gln Pro Phe Leu
165 170 175
Met Asp Leu Glu Gly Lys Gln Gly Asn Phe Lys Asn Leu Arg Glu Phe
180 185 190
Val Phe Lys Asn Ile Asp Gly Tyr Phe Lys Ile Tyr Ser Lys His Thr
195 200 205
Pro Ile Asn Leu Val Arg Asp Leu Pro Gln Gly Phe Ser Ala Leu Glu
210 215 220
Pro Leu Val Asp Leu Pro Ile Gly Ile Asn Ile Thr Arg Phe Gln Thr
225 230 235 240
Leu Leu Ala Leu His Arg Ser Tyr Leu Thr Pro Gly Asp Ser Ser Ser
245 250 255
Gly Trp Thr Ala Gly Ala Ala Ala Tyr Tyr Val Gly Tyr Leu Gln Pro
260 265 270
Arg Thr Phe Leu Leu Lys Tyr Asn Glu Asn Gly Thr Ile Thr Asp Ala
275 280 285
Val Asp Cys Ala Leu Asp Pro Leu Ser Glu Thr Lys Cys Thr Leu Lys
290 295 300
Ser Phe Thr Val Glu Lys Gly Ile Tyr Gln Thr Ser Asn Phe Arg Val
305 310 315 320
Gln Pro Ala Glu Ala Ile Val Arg Phe Pro Gln Ile Thr Asn Leu Cys
325 330 335
Pro Phe Gly Glu Val Phe Gln Ala Thr Arg Phe Ala Ser Val Tyr Ala
340 345 350
Trp Asn Arg Lys Arg Ile Ser Asn Cys Val Ala Asp Tyr Ser Val Leu
355 360 365
Tyr Asn Ser Ala Ser Phe Ser Thr Phe Lys Cys Tyr Gly Val Ser Pro
370 375 380
Thr Lys Leu Asn Asp Leu Cys Phe Thr Asn Val Tyr Ala Asp Ser Phe
385 390 395 400
Val Ile Arg Gly Asp Glu Val Arg Gln Ile Ala Pro Gly Gln Thr Gly
405 410 415
Lys Ile Ala Asp Tyr Asn Tyr Lys Leu Pro Asp Asp Phe Thr Gly Cys
420 425 430
Val Ile Ala Trp Asn Ser Asn Asn Leu Asp Ser Lys Val Gly Gly Asn
435 440 445
Tyr Asn Tyr Leu Tyr Arg Leu Phe Arg Lys Ser Asn Leu Lys Pro Phe
450 455 460
Glu Arg Asp Ile Ser Thr Glu Ile Tyr Gln Ala Gly Ser Thr Pro Cys
465 470 475 480
Asn Gly Val Glu Gly Phe Asn Cys Tyr Phe Pro Leu Gln Ser Tyr Gly
485 490 495
Phe Gln Pro Thr Asn Gly Val Gly Tyr Gln Pro Tyr Arg Val Val Val
500 505 510
Leu Ser Phe Glu Leu Leu His Ala Pro Ala Thr Val Cys Gly Pro Lys
515 520 525
Lys Ser Thr Asn Leu Val Lys Asn Lys Cys Val Asn Phe Asn Phe Asn
530 535 540
Gly Leu Thr Gly Thr Gly Val Leu Thr Glu Ser Asn Lys Lys Phe Leu
545 550 555 560
Pro Phe Gln Gln Phe Gly Arg Asp Ile Ala Asp Thr Thr Asp Ala Val
565 570 575
Arg Asp Pro Gln Thr Leu Glu Ile Leu Asp Ile Thr Pro Cys Ser Phe
580 585 590
Gly Gly Val Ser Val Ile Thr Pro Gly Thr Asn Thr Ser Asn Gln Val
595 600 605
Ala Val Leu Tyr Gln Asp Val Asn Cys Thr Glu Val Pro Val Ala Ile
610 615 620
His Ala Asp Gln Leu Thr Pro Thr Trp Arg Val Tyr Ser Thr Gly Ser
625 630 635 640
Asn Val Phe Gln Thr Arg Ala Gly Cys Leu Ile Gly Ala Glu His Val
645 650 655
Asn Asn Ser Tyr Glu Cys Asp Ile Pro Ile Gly Ala Gly Ile Cys Ala
660 665 670
Ser Tyr Gln Thr Gln Thr Asn Ser Pro Arg Arg Ala Arg Ser Val Ala
675 680 685
Ser Gln Ser Ile Ile Ala Tyr Thr Met Ser Leu Gly Ala Glu Asn Ser
690 695 700
Val Ala Tyr Ser Asn Asn Ser Ile Ala Ile Pro Thr Asn Phe Thr Ile
705 710 715 720
Ser Val Thr Thr Glu Ile Leu Pro Val Ser Met Thr Lys Thr Ser Val
725 730 735
Asp Cys Thr Met Tyr Ile Cys Gly Asp Ser Thr Glu Cys Ser Asn Leu
740 745 750
Leu Leu Gln Tyr Gly Ser Phe Cys Thr Gln Leu Asn Arg Ala Leu Thr
755 760 765
Gly Ile Ala Val Glu Gln Asp Lys Asn Thr Gln Glu Val Phe Ala Gln
770 775 780
Val Lys Gln Ile Tyr Lys Thr Pro Pro Ile Lys Asp Phe Gly Gly Phe
785 790 795 800
Gln Phe Ser Gln Ile Leu Pro Asp Pro Ser Lys Pro Ser Lys Arg Ser
805 810 815
Phe Ile Glu Asp Leu Leu Phe Asn Lys Val Thr Leu Ala Asp Ala Gly
820 825 830
Phe Ile Lys Gln Tyr Gly Asp Cys Leu Gly Asp Ile Ala Ala Arg Asp
835 840 845
Leu Ile Cys Ala Gln Lys Phe Asn Gly Leu Thr Val Leu Pro Pro Leu
850 855 860
Leu Thr Asp Glu Met Ile Ala Gln Tyr Thr Ser Ala Leu Leu Ala Gly
865 870 875 880
Thr Ile Thr Ser Gly Trp Thr Phe Gly Ala Gly Ala Ala Leu Gln Ile
885 890 895
Pro Phe Ala Met Gln Met Ala Tyr Arg Phe Asn Gly Ile Gly Val Thr
900 905 910
Gln Asn Val Leu Tyr Glu Asn Gln Lys Leu Ile Ala Asn Gln Phe Asn
915 920 925
Ser Ala Ile Gly Lys Ile Gln Asp Ser Leu Ser Ser Thr Ala Ser Ala
930 935 940
Leu Gly Lys Leu Gln Asp Val Val Asn Gln Asn Ala Gln Ala Leu Asn
945 950 955 960
Thr Leu Val Lys Gln Leu Ser Ser Asn Phe Gly Ala Ile Ser Ser Val
965 970 975
Leu Asn Asp Ile Leu Ser Arg Leu Asp Pro Pro Glu Ala Glu Val Gln
980 985 990
Ile Asp Arg Leu Ile Thr Gly Arg Leu Gln Ser Leu Gln Thr Tyr Val
995 1000 1005
Thr Gln Gln Leu Ile Arg Ala Ala Glu Ile Arg Ala Ser Ala Asn
1010 1015 1020
Leu Ala Ala Thr Lys Met Ser Glu Cys Val Leu Gly Gln Ser Lys
1025 1030 1035
Arg Val Asp Phe Cys Gly Lys Gly Tyr His Leu Met Ser Phe Pro
1040 1045 1050
Gln Ser Ala Pro His Gly Val Val Phe Leu His Val Thr Tyr Val
1055 1060 1065
Pro Ala Gln Glu Lys Asn Phe Thr Thr Ala Pro Ala Ile Cys His
1070 1075 1080
Asp Gly Lys Ala His Phe Pro Arg Glu Gly Val Phe Val Ser Asn
1085 1090 1095
Gly Thr His Trp Phe Val Thr Gln Arg Asn Phe Tyr Glu Pro Gln
1100 1105 1110
Ile Ile Thr Thr Asp Asn Thr Phe Val Ser Gly Asn Cys Asp Val
1115 1120 1125
Val Ile Gly Ile Val Asn Asn Thr Val Tyr Asp Pro Leu Gln Pro
1130 1135 1140
Glu Leu Asp Ser Phe Lys Glu Glu Leu Asp Lys Tyr Phe Lys Asn
1145 1150 1155
His Thr Ser Pro Asp Val Asp Leu Gly Asp Ile Ser Gly Ile Asn
1160 1165 1170
Ala Ser Val Val Asn Ile Gln Lys Glu Ile Asp Arg Leu Asn Glu
1175 1180 1185
Val Ala Lys Asn Leu Asn Glu Ser Leu Ile Asp Leu Gln Glu Leu
1190 1195 1200
Gly Lys Tyr Glu Gln Tyr Ile Lys Trp Pro Trp Tyr Ile Trp Leu
1205 1210 1215
Gly Phe Ile Ala Gly Leu Ile Ala Ile Val Met Val Thr Ile Met
1220 1225 1230
Leu Cys Cys Met Thr Ser Cys Cys Ser Cys Leu Lys Gly Cys Cys
1235 1240 1245
Ser Cys Gly Ser Cys Cys Lys Phe Asp Glu Asp Asp Ser Glu Pro
1250 1255 1260
Val Leu Lys Gly Val Lys Leu His Tyr Thr
1265 1270
<210> 11
<211> 3822
<212> DNA
<213> Artificial Sequence
<220>
<223> S-(deg-RBD-1194)
<400> 11
atgttcgtct tcctggtcct gctgcctctg gtctcctcac agtgcgtcaa tctgacaact 60
cggactcagc tgccacctgc ttatactaat agcttcacca gaggcgtgta ctatcctgac 120
aaggtgttta gaagctccgt gctgcactct acacaggatc tgtttctgcc attctttagc 180
aacgtgacct ggttccacgc catccacgtg agcggcacca atggcacaaa gcggttcgac 240
aatcccgtgc tgccttttaa cgatggcgtg tacttcgcct ctaccgagaa gagcaacatc 300
atcagaggct ggatctttgg caccacactg gactccaaga cacagtctct gctgatcgtg 360
aacaatgcca ccaacgtggt catcaaggtg tgcgagttcc agttttgtaa tgatcccttc 420
ctgggcgtgt actatcacaa gaacaataag agctggatgg agtccgagtt tagagtgtat 480
tctagcgcca acaactgcac atttgagtac gtgagccagc ctttcctgat ggacctggag 540
ggcaagcagg gcaatttcaa gaacctgagg gagttcgtgt ttaagaatat cgacggctac 600
ttcaaaatct actctaagca cacccccatc aacctggtgc gcgacctgcc tcagggcttc 660
agcgccctgg agcccctggt ggatctgcct atcggcatca acatcacccg gtttcagaca 720
ctgctggccc tgcacagaag ctacctgaca cccggcgact cctctagcgg atggaccgcc 780
ggcgctgccg cctactatgt gggctacctc cagccccgga ccttcctgct gaagtacaac 840
gagaatggca ccatcacaga cgcagtggat tgcgccctgg accccctgag cgagacaaag 900
tgtacactga agtcctttac cgtggagaag ggcatctatc agacatccaa tttcagggtg 960
cagccagccg aggcgatcgt gcgctttcct gaaatcacaa acctgtgccc atttggcgag 1020
gtgttcgagg caacccgctt cgccagcgtg tacgcctgga ataggaagcg gatcagcaac 1080
tgcgtggccg actatagcgt gctgtacaac tccgcctctt tcagcacctt taagtgctat 1140
ggcgtgtccc ccacaaagct gaatgacctg tgctttacca acgtctacgc cgattctttc 1200
gtgatcaggg gcgacgaggt gcgccagatc gcccccggcc agacaggcaa gatcgcagac 1260
tacaattata agctgccaga cgatttcacc ggctgcgtga tcgcctggaa cagcaacaat 1320
ctggattcca aagtgggcgg caactacaat tatctgtacc ggctgtttag aaagagcaat 1380
ctgaagccct tcgagaggga catctctaca gaaatctacc aggccggcag caccccttgc 1440
aatggcgtgg agggctttaa ctgttatttc ccactccagt cctacggctt ccagcccaca 1500
aacggcgtgg gctatcagcc ttaccgcgtg gtggtgctga gctttgagct gctgcacgcc 1560
ccagcaacag tgtgcggccc caagaagtcc accaatctgg tgaagaacaa gtgcgtgaac 1620
ttcaacttca acggcctgac cggcacaggc gtgctgaccg agtccaacaa gaagttcctg 1680
ccatttcagc agttcggcag ggacatcgca gataccacag acgccgtgcg cgacccacag 1740
accctggaga tcctggacat cacaccctgc tctttcggcg gcgtgagcgt gatcacaccc 1800
ggcaccaata caagcaacca ggtggccgtg ctgtatcagg acgtgaattg taccgaggtg 1860
cccgtggcta tccacgccga tcagctgacc ccaacatggc gggtgtacag caccggctcc 1920
aacgtcttcc agacaagagc cggatgcctg atcggagcag agcacgtgaa caattcctat 1980
gagtgcgaca tcccaatcgg cgccggcatc tgtgcctctt accagaccca gacaaactct 2040
cccagaagag cccggagcgt ggcctcccag tctatcatcg cctataccat gtccctgggc 2100
gccgagaaca gcgtggccta ctctaacaat agcatcgcca tcccaaccaa cttcacaatc 2160
tctgtgacca cagagatcct gcccgtgtcc atgaccaaga catctgtgga ctgcacaatg 2220
tatatctgtg gcgattctac cgagtgcagc aacctgctgc tccagtacgg cagcttttgt 2280
acccagctga atagagccct gacaggcatc gccgtggagc aggataagaa cacacaggag 2340
gtgttcgccc aggtgaagca aatctacaag acccccccta tcaaggactt tggcggcttc 2400
aatttttccc agatcctgcc tgatccatcc aagccttcta agcggagctt tatcgaggac 2460
ctgctgttca acaaggtgac cctggccgat gccggcttca tcaagcagta tggcgattgc 2520
ctgggcgaca tcgcagccag ggacctgatc tgcgcccaga agtttaatgg cctgaccgtg 2580
ctgccacccc tgctgacaga tgagatgatc gcacagtaca caagcgccct gctggccggc 2640
accatcacat ccggatggac cttcggcgca ggagccgccc tccagatccc ctttgccatg 2700
cagatggcct ataggttcaa cggcatcggc gtgacccaga atgtgctgta cgagaaccag 2760
aagctgatcg ccaatcagtt taactccgcc atcggcaaga tccaggacag cctgtcctct 2820
acagccagcg ccctgggcaa gctccaggat gtggtgaatc agaacgccca ggccctgaat 2880
accctggtga agcagctgag cagcaacttc ggcgccatct ctagcgtgct gaatgacatc 2940
ctgagccggc tggacaaggt ggaggcagag gtgcagatcg accggctgat caccggccgg 3000
ctccagagcc tccagaccta tgtgacacag cagctgatca gggccgccga gatcagggcc 3060
agcgccaatc tggcagcaac caagatgtcc gagtgcgtgc tgggccagtc taagagagtg 3120
gacttttgtg gcaagggcta tcacctgatg tccttccctc agtctgcccc acacggcgtg 3180
gtgtttctgc acgtgaccta cgtgcccgcc caggagaaga acttcaccac agcccctgcc 3240
atctgccacg atggcaaggc ccactttcca agggagggcg tgttcgtgtc caacggcacc 3300
cactggtttg tgacacagcg caatttctac gagccccaga tcatcaccac agacaacacc 3360
ttcgtgagcg gcaactgtga cgtggtcatc ggcatcgtga acaataccgt gtatgatcca 3420
ctccagcccg agctggacag ctttaaggag gagctggata agtatttcaa gaatcacacc 3480
tcccctgacg tggatctggg cgacatcagc ggcatcaatg cctccgtggt gaacatccag 3540
aaggagatcg accgcctgaa cgaggtggct aagaatctgc aggagagcct gatcgacctc 3600
caggagctgg gcaagtatga gcagtacatc aagtggccct ggtacatctg gctgggcttc 3660
atcgccggcc tgatcgccat cgtgatggtg accatcatgc tgtgctgtat gacatcctgc 3720
tgttcttgcc tgaagggctg ctgtagctgt ggctcctgct gtaagtttga cgaggatgac 3780
tctgaacctg tgctgaaggg cgtgaagctg cattacacct aa 3822
<210> 12
<211> 1273
<212> PRT
<213> Artificial Sequence
<220>
<223> S-(deg-RBD-1194)
<400> 12
Met Phe Val Phe Leu Val Leu Leu Pro Leu Val Ser Ser Gln Cys Val
1 5 10 15
Asn Leu Thr Thr Arg Thr Gln Leu Pro Pro Ala Tyr Thr Asn Ser Phe
20 25 30
Thr Arg Gly Val Tyr Tyr Pro Asp Lys Val Phe Arg Ser Ser Val Leu
35 40 45
His Ser Thr Gln Asp Leu Phe Leu Pro Phe Phe Ser Asn Val Thr Trp
50 55 60
Phe His Ala Ile His Val Ser Gly Thr Asn Gly Thr Lys Arg Phe Asp
65 70 75 80
Asn Pro Val Leu Pro Phe Asn Asp Gly Val Tyr Phe Ala Ser Thr Glu
85 90 95
Lys Ser Asn Ile Ile Arg Gly Trp Ile Phe Gly Thr Thr Leu Asp Ser
100 105 110
Lys Thr Gln Ser Leu Leu Ile Val Asn Asn Ala Thr Asn Val Val Ile
115 120 125
Lys Val Cys Glu Phe Gln Phe Cys Asn Asp Pro Phe Leu Gly Val Tyr
130 135 140
Tyr His Lys Asn Asn Lys Ser Trp Met Glu Ser Glu Phe Arg Val Tyr
145 150 155 160
Ser Ser Ala Asn Asn Cys Thr Phe Glu Tyr Val Ser Gln Pro Phe Leu
165 170 175
Met Asp Leu Glu Gly Lys Gln Gly Asn Phe Lys Asn Leu Arg Glu Phe
180 185 190
Val Phe Lys Asn Ile Asp Gly Tyr Phe Lys Ile Tyr Ser Lys His Thr
195 200 205
Pro Ile Asn Leu Val Arg Asp Leu Pro Gln Gly Phe Ser Ala Leu Glu
210 215 220
Pro Leu Val Asp Leu Pro Ile Gly Ile Asn Ile Thr Arg Phe Gln Thr
225 230 235 240
Leu Leu Ala Leu His Arg Ser Tyr Leu Thr Pro Gly Asp Ser Ser Ser
245 250 255
Gly Trp Thr Ala Gly Ala Ala Ala Tyr Tyr Val Gly Tyr Leu Gln Pro
260 265 270
Arg Thr Phe Leu Leu Lys Tyr Asn Glu Asn Gly Thr Ile Thr Asp Ala
275 280 285
Val Asp Cys Ala Leu Asp Pro Leu Ser Glu Thr Lys Cys Thr Leu Lys
290 295 300
Ser Phe Thr Val Glu Lys Gly Ile Tyr Gln Thr Ser Asn Phe Arg Val
305 310 315 320
Gln Pro Ala Glu Ala Ile Val Arg Phe Pro Gln Ile Thr Asn Leu Cys
325 330 335
Pro Phe Gly Glu Val Phe Gln Ala Thr Arg Phe Ala Ser Val Tyr Ala
340 345 350
Trp Asn Arg Lys Arg Ile Ser Asn Cys Val Ala Asp Tyr Ser Val Leu
355 360 365
Tyr Asn Ser Ala Ser Phe Ser Thr Phe Lys Cys Tyr Gly Val Ser Pro
370 375 380
Thr Lys Leu Asn Asp Leu Cys Phe Thr Asn Val Tyr Ala Asp Ser Phe
385 390 395 400
Val Ile Arg Gly Asp Glu Val Arg Gln Ile Ala Pro Gly Gln Thr Gly
405 410 415
Lys Ile Ala Asp Tyr Asn Tyr Lys Leu Pro Asp Asp Phe Thr Gly Cys
420 425 430
Val Ile Ala Trp Asn Ser Asn Asn Leu Asp Ser Lys Val Gly Gly Asn
435 440 445
Tyr Asn Tyr Leu Tyr Arg Leu Phe Arg Lys Ser Asn Leu Lys Pro Phe
450 455 460
Glu Arg Asp Ile Ser Thr Glu Ile Tyr Gln Ala Gly Ser Thr Pro Cys
465 470 475 480
Asn Gly Val Glu Gly Phe Asn Cys Tyr Phe Pro Leu Gln Ser Tyr Gly
485 490 495
Phe Gln Pro Thr Asn Gly Val Gly Tyr Gln Pro Tyr Arg Val Val Val
500 505 510
Leu Ser Phe Glu Leu Leu His Ala Pro Ala Thr Val Cys Gly Pro Lys
515 520 525
Lys Ser Thr Asn Leu Val Lys Asn Lys Cys Val Asn Phe Asn Phe Asn
530 535 540
Gly Leu Thr Gly Thr Gly Val Leu Thr Glu Ser Asn Lys Lys Phe Leu
545 550 555 560
Pro Phe Gln Gln Phe Gly Arg Asp Ile Ala Asp Thr Thr Asp Ala Val
565 570 575
Arg Asp Pro Gln Thr Leu Glu Ile Leu Asp Ile Thr Pro Cys Ser Phe
580 585 590
Gly Gly Val Ser Val Ile Thr Pro Gly Thr Asn Thr Ser Asn Gln Val
595 600 605
Ala Val Leu Tyr Gln Asp Val Asn Cys Thr Glu Val Pro Val Ala Ile
610 615 620
His Ala Asp Gln Leu Thr Pro Thr Trp Arg Val Tyr Ser Thr Gly Ser
625 630 635 640
Asn Val Phe Gln Thr Arg Ala Gly Cys Leu Ile Gly Ala Glu His Val
645 650 655
Asn Asn Ser Tyr Glu Cys Asp Ile Pro Ile Gly Ala Gly Ile Cys Ala
660 665 670
Ser Tyr Gln Thr Gln Thr Asn Ser Pro Arg Arg Ala Arg Ser Val Ala
675 680 685
Ser Gln Ser Ile Ile Ala Tyr Thr Met Ser Leu Gly Ala Glu Asn Ser
690 695 700
Val Ala Tyr Ser Asn Asn Ser Ile Ala Ile Pro Thr Asn Phe Thr Ile
705 710 715 720
Ser Val Thr Thr Glu Ile Leu Pro Val Ser Met Thr Lys Thr Ser Val
725 730 735
Asp Cys Thr Met Tyr Ile Cys Gly Asp Ser Thr Glu Cys Ser Asn Leu
740 745 750
Leu Leu Gln Tyr Gly Ser Phe Cys Thr Gln Leu Asn Arg Ala Leu Thr
755 760 765
Gly Ile Ala Val Glu Gln Asp Lys Asn Thr Gln Glu Val Phe Ala Gln
770 775 780
Val Lys Gln Ile Tyr Lys Thr Pro Pro Ile Lys Asp Phe Gly Gly Phe
785 790 795 800
Asn Phe Ser Gln Ile Leu Pro Asp Pro Ser Lys Pro Ser Lys Arg Ser
805 810 815
Phe Ile Glu Asp Leu Leu Phe Asn Lys Val Thr Leu Ala Asp Ala Gly
820 825 830
Phe Ile Lys Gln Tyr Gly Asp Cys Leu Gly Asp Ile Ala Ala Arg Asp
835 840 845
Leu Ile Cys Ala Gln Lys Phe Asn Gly Leu Thr Val Leu Pro Pro Leu
850 855 860
Leu Thr Asp Glu Met Ile Ala Gln Tyr Thr Ser Ala Leu Leu Ala Gly
865 870 875 880
Thr Ile Thr Ser Gly Trp Thr Phe Gly Ala Gly Ala Ala Leu Gln Ile
885 890 895
Pro Phe Ala Met Gln Met Ala Tyr Arg Phe Asn Gly Ile Gly Val Thr
900 905 910
Gln Asn Val Leu Tyr Glu Asn Gln Lys Leu Ile Ala Asn Gln Phe Asn
915 920 925
Ser Ala Ile Gly Lys Ile Gln Asp Ser Leu Ser Ser Thr Ala Ser Ala
930 935 940
Leu Gly Lys Leu Gln Asp Val Val Asn Gln Asn Ala Gln Ala Leu Asn
945 950 955 960
Thr Leu Val Lys Gln Leu Ser Ser Asn Phe Gly Ala Ile Ser Ser Val
965 970 975
Leu Asn Asp Ile Leu Ser Arg Leu Asp Pro Pro Glu Ala Glu Val Gln
980 985 990
Ile Asp Arg Leu Ile Thr Gly Arg Leu Gln Ser Leu Gln Thr Tyr Val
995 1000 1005
Thr Gln Gln Leu Ile Arg Ala Ala Glu Ile Arg Ala Ser Ala Asn
1010 1015 1020
Leu Ala Ala Thr Lys Met Ser Glu Cys Val Leu Gly Gln Ser Lys
1025 1030 1035
Arg Val Asp Phe Cys Gly Lys Gly Tyr His Leu Met Ser Phe Pro
1040 1045 1050
Gln Ser Ala Pro His Gly Val Val Phe Leu His Val Thr Tyr Val
1055 1060 1065
Pro Ala Gln Glu Lys Asn Phe Thr Thr Ala Pro Ala Ile Cys His
1070 1075 1080
Asp Gly Lys Ala His Phe Pro Arg Glu Gly Val Phe Val Ser Asn
1085 1090 1095
Gly Thr His Trp Phe Val Thr Gln Arg Asn Phe Tyr Glu Pro Gln
1100 1105 1110
Ile Ile Thr Thr Asp Asn Thr Phe Val Ser Gly Asn Cys Asp Val
1115 1120 1125
Val Ile Gly Ile Val Asn Asn Thr Val Tyr Asp Pro Leu Gln Pro
1130 1135 1140
Glu Leu Asp Ser Phe Lys Glu Glu Leu Asp Lys Tyr Phe Lys Asn
1145 1150 1155
His Thr Ser Pro Asp Val Asp Leu Gly Asp Ile Ser Gly Ile Asn
1160 1165 1170
Ala Ser Val Val Asn Ile Gln Lys Glu Ile Asp Arg Leu Asn Glu
1175 1180 1185
Val Ala Lys Asn Leu Gln Glu Ser Leu Ile Asp Leu Gln Glu Leu
1190 1195 1200
Gly Lys Tyr Glu Gln Tyr Ile Lys Trp Pro Trp Tyr Ile Trp Leu
1205 1210 1215
Gly Phe Ile Ala Gly Leu Ile Ala Ile Val Met Val Thr Ile Met
1220 1225 1230
Leu Cys Cys Met Thr Ser Cys Cys Ser Cys Leu Lys Gly Cys Cys
1235 1240 1245
Ser Cys Gly Ser Cys Cys Lys Phe Asp Glu Asp Asp Ser Glu Pro
1250 1255 1260
Val Leu Lys Gly Val Lys Leu His Tyr Thr
1265 1270
<210> 13
<211> 3822
<212> DNA
<213> Artificial Sequence
<220>
<223> S-(deg-RBD-122-165-234)
<400> 13
atgttcgtct tcctggtcct gctgcctctg gtctcctcac agtgcgtcaa tctgacaact 60
cggactcagc tgccacctgc ttatactaat agcttcacca gaggcgtgta ctatcctgac 120
aaggtgttta gaagctccgt gctgcactct acacaggatc tgtttctgcc attctttagc 180
aacgtgacct ggttccacgc catccacgtg agcggcacca atggcacaaa gcggttcgac 240
aatcccgtgc tgccttttaa cgatggcgtg tacttcgcct ctaccgagaa gagcaacatc 300
atcagaggct ggatctttgg caccacactg gactccaaga cacagtctct gctgatcgtg 360
aaccaagcca ccaacgtggt catcaaggtg tgcgagttcc agttttgtaa tgatcccttc 420
ctgggcgtgt actatcacaa gaacaataag agctggatgg agtccgagtt tagagtgtat 480
tctagcgcca accagtgcac atttgagtac gtgagccagc ctttcctgat ggacctggag 540
ggcaagcagg gcaatttcaa gaacctgagg gagttcgtgt ttaagaatat cgacggctac 600
ttcaaaatct actctaagca cacccccatc aacctggtgc gcgacctgcc tcagggcttc 660
agcgccctgg agcccctggt ggatctgcct atcggcatcc agatcacccg gtttcagaca 720
ctgctggccc tgcacagaag ctacctgaca cccggcgact cctctagcgg atggaccgcc 780
ggcgctgccg cctactatgt gggctacctc cagccccgga ccttcctgct gaagtacaac 840
gagaatggca ccatcacaga cgcagtggat tgcgccctgg accccctgag cgagacaaag 900
tgtacactga agtcctttac cgtggagaag ggcatctatc agacatccaa tttcagggtg 960
cagccagccg aggcgatcgt gcgctttcct gaaatcacaa acctgtgccc atttggcgag 1020
gtgttcgagg caacccgctt cgccagcgtg tacgcctgga ataggaagcg gatcagcaac 1080
tgcgtggccg actatagcgt gctgtacaac tccgcctctt tcagcacctt taagtgctat 1140
ggcgtgtccc ccacaaagct gaatgacctg tgctttacca acgtctacgc cgattctttc 1200
gtgatcaggg gcgacgaggt gcgccagatc gcccccggcc agacaggcaa gatcgcagac 1260
tacaattata agctgccaga cgatttcacc ggctgcgtga tcgcctggaa cagcaacaat 1320
ctggattcca aagtgggcgg caactacaat tatctgtacc ggctgtttag aaagagcaat 1380
ctgaagccct tcgagaggga catctctaca gaaatctacc aggccggcag caccccttgc 1440
aatggcgtgg agggctttaa ctgttatttc ccactccagt cctacggctt ccagcccaca 1500
aacggcgtgg gctatcagcc ttaccgcgtg gtggtgctga gctttgagct gctgcacgcc 1560
ccagcaacag tgtgcggccc caagaagtcc accaatctgg tgaagaacaa gtgcgtgaac 1620
ttcaacttca acggcctgac cggcacaggc gtgctgaccg agtccaacaa gaagttcctg 1680
ccatttcagc agttcggcag ggacatcgca gataccacag acgccgtgcg cgacccacag 1740
accctggaga tcctggacat cacaccctgc tctttcggcg gcgtgagcgt gatcacaccc 1800
ggcaccaata caagcaacca ggtggccgtg ctgtatcagg acgtgaattg taccgaggtg 1860
cccgtggcta tccacgccga tcagctgacc ccaacatggc gggtgtacag caccggctcc 1920
aacgtcttcc agacaagagc cggatgcctg atcggagcag agcacgtgaa caattcctat 1980
gagtgcgaca tcccaatcgg cgccggcatc tgtgcctctt accagaccca gacaaactct 2040
cccagaagag cccggagcgt ggcctcccag tctatcatcg cctataccat gtccctgggc 2100
gccgagaaca gcgtggccta ctctaacaat agcatcgcca tcccaaccaa cttcacaatc 2160
tctgtgacca cagagatcct gcccgtgtcc atgaccaaga catctgtgga ctgcacaatg 2220
tatatctgtg gcgattctac cgagtgcagc aacctgctgc tccagtacgg cagcttttgt 2280
acccagctga atagagccct gacaggcatc gccgtggagc aggataagaa cacacaggag 2340
gtgttcgccc aggtgaagca aatctacaag acccccccta tcaaggactt tggcggcttc 2400
aatttttccc agatcctgcc tgatccatcc aagccttcta agcggagctt tatcgaggac 2460
ctgctgttca acaaggtgac cctggccgat gccggcttca tcaagcagta tggcgattgc 2520
ctgggcgaca tcgcagccag ggacctgatc tgcgcccaga agtttaatgg cctgaccgtg 2580
ctgccacccc tgctgacaga tgagatgatc gcacagtaca caagcgccct gctggccggc 2640
accatcacat ccggatggac cttcggcgca ggagccgccc tccagatccc ctttgccatg 2700
cagatggcct ataggttcaa cggcatcggc gtgacccaga atgtgctgta cgagaaccag 2760
aagctgatcg ccaatcagtt taactccgcc atcggcaaga tccaggacag cctgtcctct 2820
acagccagcg ccctgggcaa gctccaggat gtggtgaatc agaacgccca ggccctgaat 2880
accctggtga agcagctgag cagcaacttc ggcgccatct ctagcgtgct gaatgacatc 2940
ctgagccggc tggacaaggt ggaggcagag gtgcagatcg accggctgat caccggccgg 3000
ctccagagcc tccagaccta tgtgacacag cagctgatca gggccgccga gatcagggcc 3060
agcgccaatc tggcagcaac caagatgtcc gagtgcgtgc tgggccagtc taagagagtg 3120
gacttttgtg gcaagggcta tcacctgatg tccttccctc agtctgcccc acacggcgtg 3180
gtgtttctgc acgtgaccta cgtgcccgcc caggagaaga acttcaccac agcccctgcc 3240
atctgccacg atggcaaggc ccactttcca agggagggcg tgttcgtgtc caacggcacc 3300
cactggtttg tgacacagcg caatttctac gagccccaga tcatcaccac agacaacacc 3360
ttcgtgagcg gcaactgtga cgtggtcatc ggcatcgtga acaataccgt gtatgatcca 3420
ctccagcccg agctggacag ctttaaggag gagctggata agtatttcaa gaatcacacc 3480
tcccctgacg tggatctggg cgacatcagc ggcatcaatg cctccgtggt gaacatccag 3540
aaggagatcg accgcctgaa cgaggtggct aagaatctga acgagagcct gatcgacctc 3600
caggagctgg gcaagtatga gcagtacatc aagtggccct ggtacatctg gctgggcttc 3660
atcgccggcc tgatcgccat cgtgatggtg accatcatgc tgtgctgtat gacatcctgc 3720
tgttcttgcc tgaagggctg ctgtagctgt ggctcctgct gtaagtttga cgaggatgac 3780
tctgaacctg tgctgaaggg cgtgaagctg cattacacct aa 3822
<210> 14
<211> 1273
<212> PRT
<213> Artificial Sequence
<220>
<223> S-(deg-RBD-122-165-234)
<400> 14
Met Phe Val Phe Leu Val Leu Leu Pro Leu Val Ser Ser Gln Cys Val
1 5 10 15
Asn Leu Thr Thr Arg Thr Gln Leu Pro Pro Ala Tyr Thr Asn Ser Phe
20 25 30
Thr Arg Gly Val Tyr Tyr Pro Asp Lys Val Phe Arg Ser Ser Val Leu
35 40 45
His Ser Thr Gln Asp Leu Phe Leu Pro Phe Phe Ser Asn Val Thr Trp
50 55 60
Phe His Ala Ile His Val Ser Gly Thr Asn Gly Thr Lys Arg Phe Asp
65 70 75 80
Asn Pro Val Leu Pro Phe Asn Asp Gly Val Tyr Phe Ala Ser Thr Glu
85 90 95
Lys Ser Asn Ile Ile Arg Gly Trp Ile Phe Gly Thr Thr Leu Asp Ser
100 105 110
Lys Thr Gln Ser Leu Leu Ile Val Asn Gln Ala Thr Asn Val Val Ile
115 120 125
Lys Val Cys Glu Phe Gln Phe Cys Asn Asp Pro Phe Leu Gly Val Tyr
130 135 140
Tyr His Lys Asn Asn Lys Ser Trp Met Glu Ser Glu Phe Arg Val Tyr
145 150 155 160
Ser Ser Ala Asn Gln Cys Thr Phe Glu Tyr Val Ser Gln Pro Phe Leu
165 170 175
Met Asp Leu Glu Gly Lys Gln Gly Asn Phe Lys Asn Leu Arg Glu Phe
180 185 190
Val Phe Lys Asn Ile Asp Gly Tyr Phe Lys Ile Tyr Ser Lys His Thr
195 200 205
Pro Ile Asn Leu Val Arg Asp Leu Pro Gln Gly Phe Ser Ala Leu Glu
210 215 220
Pro Leu Val Asp Leu Pro Ile Gly Ile Gln Ile Thr Arg Phe Gln Thr
225 230 235 240
Leu Leu Ala Leu His Arg Ser Tyr Leu Thr Pro Gly Asp Ser Ser Ser
245 250 255
Gly Trp Thr Ala Gly Ala Ala Ala Tyr Tyr Val Gly Tyr Leu Gln Pro
260 265 270
Arg Thr Phe Leu Leu Lys Tyr Asn Glu Asn Gly Thr Ile Thr Asp Ala
275 280 285
Val Asp Cys Ala Leu Asp Pro Leu Ser Glu Thr Lys Cys Thr Leu Lys
290 295 300
Ser Phe Thr Val Glu Lys Gly Ile Tyr Gln Thr Ser Asn Phe Arg Val
305 310 315 320
Gln Pro Ala Glu Ala Ile Val Arg Phe Pro Gln Ile Thr Asn Leu Cys
325 330 335
Pro Phe Gly Glu Val Phe Gln Ala Thr Arg Phe Ala Ser Val Tyr Ala
340 345 350
Trp Asn Arg Lys Arg Ile Ser Asn Cys Val Ala Asp Tyr Ser Val Leu
355 360 365
Tyr Asn Ser Ala Ser Phe Ser Thr Phe Lys Cys Tyr Gly Val Ser Pro
370 375 380
Thr Lys Leu Asn Asp Leu Cys Phe Thr Asn Val Tyr Ala Asp Ser Phe
385 390 395 400
Val Ile Arg Gly Asp Glu Val Arg Gln Ile Ala Pro Gly Gln Thr Gly
405 410 415
Lys Ile Ala Asp Tyr Asn Tyr Lys Leu Pro Asp Asp Phe Thr Gly Cys
420 425 430
Val Ile Ala Trp Asn Ser Asn Asn Leu Asp Ser Lys Val Gly Gly Asn
435 440 445
Tyr Asn Tyr Leu Tyr Arg Leu Phe Arg Lys Ser Asn Leu Lys Pro Phe
450 455 460
Glu Arg Asp Ile Ser Thr Glu Ile Tyr Gln Ala Gly Ser Thr Pro Cys
465 470 475 480
Asn Gly Val Glu Gly Phe Asn Cys Tyr Phe Pro Leu Gln Ser Tyr Gly
485 490 495
Phe Gln Pro Thr Asn Gly Val Gly Tyr Gln Pro Tyr Arg Val Val Val
500 505 510
Leu Ser Phe Glu Leu Leu His Ala Pro Ala Thr Val Cys Gly Pro Lys
515 520 525
Lys Ser Thr Asn Leu Val Lys Asn Lys Cys Val Asn Phe Asn Phe Asn
530 535 540
Gly Leu Thr Gly Thr Gly Val Leu Thr Glu Ser Asn Lys Lys Phe Leu
545 550 555 560
Pro Phe Gln Gln Phe Gly Arg Asp Ile Ala Asp Thr Thr Asp Ala Val
565 570 575
Arg Asp Pro Gln Thr Leu Glu Ile Leu Asp Ile Thr Pro Cys Ser Phe
580 585 590
Gly Gly Val Ser Val Ile Thr Pro Gly Thr Asn Thr Ser Asn Gln Val
595 600 605
Ala Val Leu Tyr Gln Asp Val Asn Cys Thr Glu Val Pro Val Ala Ile
610 615 620
His Ala Asp Gln Leu Thr Pro Thr Trp Arg Val Tyr Ser Thr Gly Ser
625 630 635 640
Asn Val Phe Gln Thr Arg Ala Gly Cys Leu Ile Gly Ala Glu His Val
645 650 655
Asn Asn Ser Tyr Glu Cys Asp Ile Pro Ile Gly Ala Gly Ile Cys Ala
660 665 670
Ser Tyr Gln Thr Gln Thr Asn Ser Pro Arg Arg Ala Arg Ser Val Ala
675 680 685
Ser Gln Ser Ile Ile Ala Tyr Thr Met Ser Leu Gly Ala Glu Asn Ser
690 695 700
Val Ala Tyr Ser Asn Asn Ser Ile Ala Ile Pro Thr Asn Phe Thr Ile
705 710 715 720
Ser Val Thr Thr Glu Ile Leu Pro Val Ser Met Thr Lys Thr Ser Val
725 730 735
Asp Cys Thr Met Tyr Ile Cys Gly Asp Ser Thr Glu Cys Ser Asn Leu
740 745 750
Leu Leu Gln Tyr Gly Ser Phe Cys Thr Gln Leu Asn Arg Ala Leu Thr
755 760 765
Gly Ile Ala Val Glu Gln Asp Lys Asn Thr Gln Glu Val Phe Ala Gln
770 775 780
Val Lys Gln Ile Tyr Lys Thr Pro Pro Ile Lys Asp Phe Gly Gly Phe
785 790 795 800
Asn Phe Ser Gln Ile Leu Pro Asp Pro Ser Lys Pro Ser Lys Arg Ser
805 810 815
Phe Ile Glu Asp Leu Leu Phe Asn Lys Val Thr Leu Ala Asp Ala Gly
820 825 830
Phe Ile Lys Gln Tyr Gly Asp Cys Leu Gly Asp Ile Ala Ala Arg Asp
835 840 845
Leu Ile Cys Ala Gln Lys Phe Asn Gly Leu Thr Val Leu Pro Pro Leu
850 855 860
Leu Thr Asp Glu Met Ile Ala Gln Tyr Thr Ser Ala Leu Leu Ala Gly
865 870 875 880
Thr Ile Thr Ser Gly Trp Thr Phe Gly Ala Gly Ala Ala Leu Gln Ile
885 890 895
Pro Phe Ala Met Gln Met Ala Tyr Arg Phe Asn Gly Ile Gly Val Thr
900 905 910
Gln Asn Val Leu Tyr Glu Asn Gln Lys Leu Ile Ala Asn Gln Phe Asn
915 920 925
Ser Ala Ile Gly Lys Ile Gln Asp Ser Leu Ser Ser Thr Ala Ser Ala
930 935 940
Leu Gly Lys Leu Gln Asp Val Val Asn Gln Asn Ala Gln Ala Leu Asn
945 950 955 960
Thr Leu Val Lys Gln Leu Ser Ser Asn Phe Gly Ala Ile Ser Ser Val
965 970 975
Leu Asn Asp Ile Leu Ser Arg Leu Asp Pro Pro Glu Ala Glu Val Gln
980 985 990
Ile Asp Arg Leu Ile Thr Gly Arg Leu Gln Ser Leu Gln Thr Tyr Val
995 1000 1005
Thr Gln Gln Leu Ile Arg Ala Ala Glu Ile Arg Ala Ser Ala Asn
1010 1015 1020
Leu Ala Ala Thr Lys Met Ser Glu Cys Val Leu Gly Gln Ser Lys
1025 1030 1035
Arg Val Asp Phe Cys Gly Lys Gly Tyr His Leu Met Ser Phe Pro
1040 1045 1050
Gln Ser Ala Pro His Gly Val Val Phe Leu His Val Thr Tyr Val
1055 1060 1065
Pro Ala Gln Glu Lys Asn Phe Thr Thr Ala Pro Ala Ile Cys His
1070 1075 1080
Asp Gly Lys Ala His Phe Pro Arg Glu Gly Val Phe Val Ser Asn
1085 1090 1095
Gly Thr His Trp Phe Val Thr Gln Arg Asn Phe Tyr Glu Pro Gln
1100 1105 1110
Ile Ile Thr Thr Asp Asn Thr Phe Val Ser Gly Asn Cys Asp Val
1115 1120 1125
Val Ile Gly Ile Val Asn Asn Thr Val Tyr Asp Pro Leu Gln Pro
1130 1135 1140
Glu Leu Asp Ser Phe Lys Glu Glu Leu Asp Lys Tyr Phe Lys Asn
1145 1150 1155
His Thr Ser Pro Asp Val Asp Leu Gly Asp Ile Ser Gly Ile Asn
1160 1165 1170
Ala Ser Val Val Asn Ile Gln Lys Glu Ile Asp Arg Leu Asn Glu
1175 1180 1185
Val Ala Lys Asn Leu Asn Glu Ser Leu Ile Asp Leu Gln Glu Leu
1190 1195 1200
Gly Lys Tyr Glu Gln Tyr Ile Lys Trp Pro Trp Tyr Ile Trp Leu
1205 1210 1215
Gly Phe Ile Ala Gly Leu Ile Ala Ile Val Met Val Thr Ile Met
1220 1225 1230
Leu Cys Cys Met Thr Ser Cys Cys Ser Cys Leu Lys Gly Cys Cys
1235 1240 1245
Ser Cys Gly Ser Cys Cys Lys Phe Asp Glu Asp Asp Ser Glu Pro
1250 1255 1260
Val Leu Lys Gly Val Lys Leu His Tyr Thr
1265 1270
<210> 15
<211> 3816
<212> DNA
<213> SARS-CoV-2 (Delta strain)
<400> 15
atgttcgtct tcctggtcct gctgcctctg gtctcctcac agtgcgtcaa tctgcgaact 60
cggactcagc tgccacctgc ttatactaat agcttcacca gaggcgtgta ctatcctgac 120
aaggtgttta gaagctccgt gctgcactct acacaggatc tgtttctgcc attctttagc 180
aacgtgacct ggttccacgc catccacgtg agcggcacca atggcacaaa gcggttcgac 240
aatcccgtgc tgccttttaa cgatggcgtg tacttcgcct ctatcgagaa gagcaacatc 300
atcagaggct ggatctttgg caccacactg gactccaaga cacagtctct gctgatcgtg 360
aacaatgcca ccaacgtggt catcaaggtg tgcgagttcc agttttgtaa tgatcccttc 420
ctggacgtgt actatcacaa gaacaataag agctggatgg agtccggagt gtattctagc 480
gccaacaact gcacatttga gtacgtgagc cagcctttcc tgatggacct ggagggcaag 540
cagggcaatt tcaagaacct gagggagttc gtgtttaaga atatcgacgg ctacttcaaa 600
atctactcta agcacacccc catcaacctg gtgcgcgacc tgcctcaggg cttcagcgcc 660
ctggagcccc tggtggatct gcctatcggc atcaacatca cccggtttca gacactgctg 720
gccctgcaca gaagctacct gacacccggc gactcctcta gcggatggac cgccggcgct 780
gccgcctact atgtgggcta cctccagccc cggaccttcc tgctgaagta caacgagaat 840
ggcaccatca cagacgcagt ggattgcgcc ctggaccccc tgagcgagac aaagtgtaca 900
ctgaagtcct ttaccgtgga gaagggcatc tatcagacat ccaatttcag ggtgcagcca 960
accgagtcta tcgtgcgctt tcctaatatc acaaacctgt gcccatttgg cgaggtgttc 1020
aacgcaaccc gcttcgccag cgtgtacgcc tggaatagga agcggatcag caactgcgtg 1080
gccgactata gcgtgctgta caactccgcc tctttcagca cctttaagtg ctatggcgtg 1140
tcccccacaa agctgaatga cctgtgcttt accaacgtct acgccgattc tttcgtgatc 1200
aggggcgacg aggtgcgcca gatcgccccc ggccagacag gcaagatcgc agactacaat 1260
tataagctgc cagacgattt caccggctgc gtgatcgcct ggaacagcaa caatctggat 1320
tccaaagtgg gcggcaacta caattatcgg taccggctgt ttagaaagag caatctgaag 1380
cccttcgaga gggacatctc tacagaaatc taccaggccg gcagcaagcc ttgcaatggc 1440
gtggagggct ttaactgtta tttcccactc cagtcctacg gcttccagcc cacaaacggc 1500
gtgggctatc agccttaccg cgtggtggtg ctgagctttg agctgctgca cgccccagca 1560
acagtgtgcg gccccaagaa gtccaccaat ctggtgaaga acaagtgcgt gaacttcaac 1620
ttcaacggcc tgaccggcac aggcgtgctg accgagtcca acaagaagtt cctgccattt 1680
cagcagttcg gcagggacat cgcagatacc acagacgccg tgcgcgaccc acagaccctg 1740
gagatcctgg acatcacacc ctgctctttc ggcggcgtga gcgtgatcac acccggcacc 1800
aatacaagca accaggtggc cgtgctgtat cagggcgtga attgtaccga ggtgcccgtg 1860
gctatccacg ccgatcagct gaccccaaca tggcgggtgt acagcaccgg ctccaacgtc 1920
ttccagacaa gagccggatg cctgatcgga gcagagcacg tgaacaattc ctatgagtgc 1980
gacatcccaa tcggcgccgg catctgtgcc tcttaccaga cccagacaaa ctctcgcaga 2040
agagcccgga gcgtggcctc ccagtctatc atcgcctata ccatgtccct gggcgccgag 2100
aacagcgtgg cctactctaa caatagcatc gccatcccaa ccaacttcac aatctctgtg 2160
accacagaga tcctgcccgt gtccatgacc aagacatctg tggactgcac aatgtatatc 2220
tgtggcgatt ctaccgagtg cagcaacctg ctgctccagt acggcagctt ttgtacccag 2280
ctgaatagag ccctgacagg catcgccgtg gagcaggata agaacacaca ggaggtgttc 2340
gcccaggtga agcaaatcta caagaccccc cctatcaagg actttggcgg cttcaatttt 2400
tcccagatcc tgcctgatcc atccaagcct tctaagcgga gctttatcga ggacctgctg 2460
ttcaacaagg tgaccctggc cgatgccggc ttcatcaagc agtatggcga ttgcctgggc 2520
gacatcgcag ccagggacct gatctgcgcc cagaagttta atggcctgac cgtgctgcca 2580
cccctgctga cagatgagat gatcgcacag tacacaagcg ccctgctggc cggcaccatc 2640
acatccggat ggaccttcgg cgcaggagcc gccctccaga tcccctttgc catgcagatg 2700
gcctataggt tcaacggcat cggcgtgacc cagaatgtgc tgtacgagaa ccagaagctg 2760
atcgccaatc agtttaactc cgccatcggc aagatccagg acagcctgtc ctctacagcc 2820
agcgccctgg gcaagctcca gaatgtggtg aatcagaacg cccaggccct gaataccctg 2880
gtgaagcagc tgagcagcaa cttcggcgcc atctctagcg tgctgaatga catcctgagc 2940
cggctggaca aggtggaggc agaggtgcag atcgaccggc tgatcaccgg ccggctccag 3000
agcctccaga cctatgtgac acagcagctg atcagggccg ccgagatcag ggccagcgcc 3060
aatctggcag caaccaagat gtccgagtgc gtgctgggcc agtctaagag agtggacttt 3120
tgtggcaagg gctatcacct gatgtccttc cctcagtctg ccccacacgg cgtggtgttt 3180
ctgcacgtga cctacgtgcc cgcccaggag aagaacttca ccacagcccc tgccatctgc 3240
cacgatggca aggcccactt tccaagggag ggcgtgttcg tgtccaacgg cacccactgg 3300
tttgtgacac agcgcaattt ctacgagccc cagatcatca ccacagacaa caccttcgtg 3360
agcggcaact gtgacgtggt catcggcatc gtgaacaata ccgtgtatga tccactccag 3420
cccgagctgg acagctttaa ggaggagctg gataagtatt tcaagaatca cacctcccct 3480
gacgtggatc tgggcgacat cagcggcatc aatgcctccg tggtgaacat ccagaaggag 3540
atcgaccgcc tgaacgaggt ggctaagaat ctgaacgaga gcctgatcga cctccaggag 3600
ctgggcaagt atgagcagta catcaagtgg ccctggtaca tctggctggg cttcatcgcc 3660
ggcctgatcg ccatcgtgat ggtgaccatc atgctgtgct gtatgacatc ctgctgttct 3720
tgcctgaagg gctgctgtag ctgtggctcc tgctgtaagt ttgacgagga tgactctgaa 3780
cctgtgctga agggcgtgaa gctgcattac acctaa 3816
<210> 16
<211> 1271
<212> PRT
<213> SARS-CoV-2 (Delta strain)
<400> 16
Met Phe Val Phe Leu Val Leu Leu Pro Leu Val Ser Ser Gln Cys Val
1 5 10 15
Asn Leu Arg Thr Arg Thr Gln Leu Pro Pro Ala Tyr Thr Asn Ser Phe
20 25 30
Thr Arg Gly Val Tyr Tyr Pro Asp Lys Val Phe Arg Ser Ser Val Leu
35 40 45
His Ser Thr Gln Asp Leu Phe Leu Pro Phe Phe Ser Asn Val Thr Trp
50 55 60
Phe His Ala Ile His Val Ser Gly Thr Asn Gly Thr Lys Arg Phe Asp
65 70 75 80
Asn Pro Val Leu Pro Phe Asn Asp Gly Val Tyr Phe Ala Ser Ile Glu
85 90 95
Lys Ser Asn Ile Ile Arg Gly Trp Ile Phe Gly Thr Thr Leu Asp Ser
100 105 110
Lys Thr Gln Ser Leu Leu Ile Val Asn Asn Ala Thr Asn Val Val Ile
115 120 125
Lys Val Cys Glu Phe Gln Phe Cys Asn Asp Pro Phe Leu Asp Val Tyr
130 135 140
Tyr His Lys Asn Asn Lys Ser Trp Met Glu Ser Gly Val Tyr Ser Ser
145 150 155 160
Ala Asn Asn Cys Thr Phe Glu Tyr Val Ser Gln Pro Phe Leu Met Asp
165 170 175
Leu Glu Gly Lys Gln Gly Asn Phe Lys Asn Leu Arg Glu Phe Val Phe
180 185 190
Lys Asn Ile Asp Gly Tyr Phe Lys Ile Tyr Ser Lys His Thr Pro Ile
195 200 205
Asn Leu Val Arg Asp Leu Pro Gln Gly Phe Ser Ala Leu Glu Pro Leu
210 215 220
Val Asp Leu Pro Ile Gly Ile Asn Ile Thr Arg Phe Gln Thr Leu Leu
225 230 235 240
Ala Leu His Arg Ser Tyr Leu Thr Pro Gly Asp Ser Ser Ser Gly Trp
245 250 255
Thr Ala Gly Ala Ala Ala Tyr Tyr Val Gly Tyr Leu Gln Pro Arg Thr
260 265 270
Phe Leu Leu Lys Tyr Asn Glu Asn Gly Thr Ile Thr Asp Ala Val Asp
275 280 285
Cys Ala Leu Asp Pro Leu Ser Glu Thr Lys Cys Thr Leu Lys Ser Phe
290 295 300
Thr Val Glu Lys Gly Ile Tyr Gln Thr Ser Asn Phe Arg Val Gln Pro
305 310 315 320
Thr Glu Ser Ile Val Arg Phe Pro Asn Ile Thr Asn Leu Cys Pro Phe
325 330 335
Gly Glu Val Phe Asn Ala Thr Arg Phe Ala Ser Val Tyr Ala Trp Asn
340 345 350
Arg Lys Arg Ile Ser Asn Cys Val Ala Asp Tyr Ser Val Leu Tyr Asn
355 360 365
Ser Ala Ser Phe Ser Thr Phe Lys Cys Tyr Gly Val Ser Pro Thr Lys
370 375 380
Leu Asn Asp Leu Cys Phe Thr Asn Val Tyr Ala Asp Ser Phe Val Ile
385 390 395 400
Arg Gly Asp Glu Val Arg Gln Ile Ala Pro Gly Gln Thr Gly Lys Ile
405 410 415
Ala Asp Tyr Asn Tyr Lys Leu Pro Asp Asp Phe Thr Gly Cys Val Ile
420 425 430
Ala Trp Asn Ser Asn Asn Leu Asp Ser Lys Val Gly Gly Asn Tyr Asn
435 440 445
Tyr Arg Tyr Arg Leu Phe Arg Lys Ser Asn Leu Lys Pro Phe Glu Arg
450 455 460
Asp Ile Ser Thr Glu Ile Tyr Gln Ala Gly Ser Lys Pro Cys Asn Gly
465 470 475 480
Val Glu Gly Phe Asn Cys Tyr Phe Pro Leu Gln Ser Tyr Gly Phe Gln
485 490 495
Pro Thr Asn Gly Val Gly Tyr Gln Pro Tyr Arg Val Val Val Leu Ser
500 505 510
Phe Glu Leu Leu His Ala Pro Ala Thr Val Cys Gly Pro Lys Lys Ser
515 520 525
Thr Asn Leu Val Lys Asn Lys Cys Val Asn Phe Asn Phe Asn Gly Leu
530 535 540
Thr Gly Thr Gly Val Leu Thr Glu Ser Asn Lys Lys Phe Leu Pro Phe
545 550 555 560
Gln Gln Phe Gly Arg Asp Ile Ala Asp Thr Thr Asp Ala Val Arg Asp
565 570 575
Pro Gln Thr Leu Glu Ile Leu Asp Ile Thr Pro Cys Ser Phe Gly Gly
580 585 590
Val Ser Val Ile Thr Pro Gly Thr Asn Thr Ser Asn Gln Val Ala Val
595 600 605
Leu Tyr Gln Gly Val Asn Cys Thr Glu Val Pro Val Ala Ile His Ala
610 615 620
Asp Gln Leu Thr Pro Thr Trp Arg Val Tyr Ser Thr Gly Ser Asn Val
625 630 635 640
Phe Gln Thr Arg Ala Gly Cys Leu Ile Gly Ala Glu His Val Asn Asn
645 650 655
Ser Tyr Glu Cys Asp Ile Pro Ile Gly Ala Gly Ile Cys Ala Ser Tyr
660 665 670
Gln Thr Gln Thr Asn Ser Arg Arg Arg Ala Arg Ser Val Ala Ser Gln
675 680 685
Ser Ile Ile Ala Tyr Thr Met Ser Leu Gly Ala Glu Asn Ser Val Ala
690 695 700
Tyr Ser Asn Asn Ser Ile Ala Ile Pro Thr Asn Phe Thr Ile Ser Val
705 710 715 720
Thr Thr Glu Ile Leu Pro Val Ser Met Thr Lys Thr Ser Val Asp Cys
725 730 735
Thr Met Tyr Ile Cys Gly Asp Ser Thr Glu Cys Ser Asn Leu Leu Leu
740 745 750
Gln Tyr Gly Ser Phe Cys Thr Gln Leu Asn Arg Ala Leu Thr Gly Ile
755 760 765
Ala Val Glu Gln Asp Lys Asn Thr Gln Glu Val Phe Ala Gln Val Lys
770 775 780
Gln Ile Tyr Lys Thr Pro Pro Ile Lys Asp Phe Gly Gly Phe Asn Phe
785 790 795 800
Ser Gln Ile Leu Pro Asp Pro Ser Lys Pro Ser Lys Arg Ser Phe Ile
805 810 815
Glu Asp Leu Leu Phe Asn Lys Val Thr Leu Ala Asp Ala Gly Phe Ile
820 825 830
Lys Gln Tyr Gly Asp Cys Leu Gly Asp Ile Ala Ala Arg Asp Leu Ile
835 840 845
Cys Ala Gln Lys Phe Asn Gly Leu Thr Val Leu Pro Pro Leu Leu Thr
850 855 860
Asp Glu Met Ile Ala Gln Tyr Thr Ser Ala Leu Leu Ala Gly Thr Ile
865 870 875 880
Thr Ser Gly Trp Thr Phe Gly Ala Gly Ala Ala Leu Gln Ile Pro Phe
885 890 895
Ala Met Gln Met Ala Tyr Arg Phe Asn Gly Ile Gly Val Thr Gln Asn
900 905 910
Val Leu Tyr Glu Asn Gln Lys Leu Ile Ala Asn Gln Phe Asn Ser Ala
915 920 925
Ile Gly Lys Ile Gln Asp Ser Leu Ser Ser Thr Ala Ser Ala Leu Gly
930 935 940
Lys Leu Gln Asn Val Val Asn Gln Asn Ala Gln Ala Leu Asn Thr Leu
945 950 955 960
Val Lys Gln Leu Ser Ser Asn Phe Gly Ala Ile Ser Ser Val Leu Asn
965 970 975
Asp Ile Leu Ser Arg Leu Asp Lys Val Glu Ala Glu Val Gln Ile Asp
980 985 990
Arg Leu Ile Thr Gly Arg Leu Gln Ser Leu Gln Thr Tyr Val Thr Gln
995 1000 1005
Gln Leu Ile Arg Ala Ala Glu Ile Arg Ala Ser Ala Asn Leu Ala
1010 1015 1020
Ala Thr Lys Met Ser Glu Cys Val Leu Gly Gln Ser Lys Arg Val
1025 1030 1035
Asp Phe Cys Gly Lys Gly Tyr His Leu Met Ser Phe Pro Gln Ser
1040 1045 1050
Ala Pro His Gly Val Val Phe Leu His Val Thr Tyr Val Pro Ala
1055 1060 1065
Gln Glu Lys Asn Phe Thr Thr Ala Pro Ala Ile Cys His Asp Gly
1070 1075 1080
Lys Ala His Phe Pro Arg Glu Gly Val Phe Val Ser Asn Gly Thr
1085 1090 1095
His Trp Phe Val Thr Gln Arg Asn Phe Tyr Glu Pro Gln Ile Ile
1100 1105 1110
Thr Thr Asp Asn Thr Phe Val Ser Gly Asn Cys Asp Val Val Ile
1115 1120 1125
Gly Ile Val Asn Asn Thr Val Tyr Asp Pro Leu Gln Pro Glu Leu
1130 1135 1140
Asp Ser Phe Lys Glu Glu Leu Asp Lys Tyr Phe Lys Asn His Thr
1145 1150 1155
Ser Pro Asp Val Asp Leu Gly Asp Ile Ser Gly Ile Asn Ala Ser
1160 1165 1170
Val Val Asn Ile Gln Lys Glu Ile Asp Arg Leu Asn Glu Val Ala
1175 1180 1185
Lys Asn Leu Asn Glu Ser Leu Ile Asp Leu Gln Glu Leu Gly Lys
1190 1195 1200
Tyr Glu Gln Tyr Ile Lys Trp Pro Trp Tyr Ile Trp Leu Gly Phe
1205 1210 1215
Ile Ala Gly Leu Ile Ala Ile Val Met Val Thr Ile Met Leu Cys
1220 1225 1230
Cys Met Thr Ser Cys Cys Ser Cys Leu Lys Gly Cys Cys Ser Cys
1235 1240 1245
Gly Ser Cys Cys Lys Phe Asp Glu Asp Asp Ser Glu Pro Val Leu
1250 1255 1260
Lys Gly Val Lys Leu His Tyr Thr
1265 1270
<210> 17
<211> 3822
<212> DNA
<213> SARS-CoV-2 (Wuhan strain S-2P strain)
<400> 17
atgttcgtct tcctggtcct gctgcctctg gtctcctcac agtgcgtcaa tctgacaact 60
cggactcagc tgccacctgc ttatactaat agcttcacca gaggcgtgta ctatcctgac 120
aaggtgttta gaagctccgt gctgcactct acacaggatc tgtttctgcc attctttagc 180
aacgtgacct ggttccacgc catccacgtg agcggcacca atggcacaaa gcggttcgac 240
aatcccgtgc tgccttttaa cgatggcgtg tacttcgcct ctaccgagaa gagcaacatc 300
atcagaggct ggatctttgg caccacactg gactccaaga cacagtctct gctgatcgtg 360
aacaatgcca ccaacgtggt catcaaggtg tgcgagttcc agttttgtaa tgatcccttc 420
ctgggcgtgt actatcacaa gaacaataag agctggatgg agtccgagtt tagagtgtat 480
tctagcgcca acaactgcac atttgagtac gtgagccagc ctttcctgat ggacctggag 540
ggcaagcagg gcaatttcaa gaacctgagg gagttcgtgt ttaagaatat cgacggctac 600
ttcaaaatct actctaagca cacccccatc aacctggtgc gcgacctgcc tcagggcttc 660
agcgccctgg agcccctggt ggatctgcct atcggcatca acatcacccg gtttcagaca 720
ctgctggccc tgcacagaag ctacctgaca cccggcgact cctctagcgg atggaccgcc 780
ggcgctgccg cctactatgt gggctacctc cagccccgga ccttcctgct gaagtacaac 840
gagaatggca ccatcacaga cgcagtggat tgcgccctgg accccctgag cgagacaaag 900
tgtacactga agtcctttac cgtggagaag ggcatctatc agacatccaa tttcagggtg 960
cagccaaccg agtctatcgt gcgctttcct aatatcacaa acctgtgccc atttggcgag 1020
gtgttcaacg caacccgctt cgccagcgtg tacgcctgga ataggaagcg gatcagcaac 1080
tgcgtggccg actatagcgt gctgtacaac tccgcctctt tcagcacctt taagtgctat 1140
ggcgtgtccc ccacaaagct gaatgacctg tgctttacca acgtctacgc cgattctttc 1200
gtgatcaggg gcgacgaggt gcgccagatc gcccccggcc agacaggcaa gatcgcagac 1260
tacaattata agctgccaga cgatttcacc ggctgcgtga tcgcctggaa cagcaacaat 1320
ctggattcca aagtgggcgg caactacaat tatctgtacc ggctgtttag aaagagcaat 1380
ctgaagccct tcgagaggga catctctaca gaaatctacc aggccggcag caccccttgc 1440
aatggcgtgg agggctttaa ctgttatttc ccactccagt cctacggctt ccagcccaca 1500
aacggcgtgg gctatcagcc ttaccgcgtg gtggtgctga gctttgagct gctgcacgcc 1560
ccagcaacag tgtgcggccc caagaagtcc accaatctgg tgaagaacaa gtgcgtgaac 1620
ttcaacttca acggcctgac cggcacaggc gtgctgaccg agtccaacaa gaagttcctg 1680
ccatttcagc agttcggcag ggacatcgca gataccacag acgccgtgcg cgacccacag 1740
accctggaga tcctggacat cacaccctgc tctttcggcg gcgtgagcgt gatcacaccc 1800
ggcaccaata caagcaacca ggtggccgtg ctgtatcagg acgtgaattg taccgaggtg 1860
cccgtggcta tccacgccga tcagctgacc ccaacatggc gggtgtacag caccggctcc 1920
aacgtcttcc agacaagagc cggatgcctg atcggagcag agcacgtgaa caattcctat 1980
gagtgcgaca tcccaatcgg cgccggcatc tgtgcctctt accagaccca gacaaactct 2040
cccagaagag cccggagcgt ggcctcccag tctatcatcg cctataccat gtccctgggc 2100
gccgagaaca gcgtggccta ctctaacaat agcatcgcca tcccaaccaa cttcacaatc 2160
tctgtgacca cagagatcct gcccgtgtcc atgaccaaga catctgtgga ctgcacaatg 2220
tatatctgtg gcgattctac cgagtgcagc aacctgctgc tccagtacgg cagcttttgt 2280
acccagctga atagagccct gacaggcatc gccgtggagc aggataagaa cacacaggag 2340
gtgttcgccc aggtgaagca aatctacaag acccccccta tcaaggactt tggcggcttc 2400
aatttttccc agatcctgcc tgatccatcc aagccttcta agcggagctt tatcgaggac 2460
ctgctgttca acaaggtgac cctggccgat gccggcttca tcaagcagta tggcgattgc 2520
ctgggcgaca tcgcagccag ggacctgatc tgcgcccaga agtttaatgg cctgaccgtg 2580
ctgccacccc tgctgacaga tgagatgatc gcacagtaca caagcgccct gctggccggc 2640
accatcacat ccggatggac cttcggcgca ggagccgccc tccagatccc ctttgccatg 2700
cagatggcct ataggttcaa cggcatcggc gtgacccaga atgtgctgta cgagaaccag 2760
aagctgatcg ccaatcagtt taactccgcc atcggcaaga tccaggacag cctgtcctct 2820
acagccagcg ccctgggcaa gctccaggat gtggtgaatc agaacgccca ggccctgaat 2880
accctggtga agcagctgag cagcaacttc ggcgccatct ctagcgtgct gaatgacatc 2940
ctgagccggc tggacccgcc ggaggcagag gtgcagatcg accggctgat caccggccgg 3000
ctccagagcc tccagaccta tgtgacacag cagctgatca gggccgccga gatcagggcc 3060
agcgccaatc tggcagcaac caagatgtcc gagtgcgtgc tgggccagtc taagagagtg 3120
gacttttgtg gcaagggcta tcacctgatg tccttccctc agtctgcccc acacggcgtg 3180
gtgtttctgc acgtgaccta cgtgcccgcc caggagaaga acttcaccac agcccctgcc 3240
atctgccacg atggcaaggc ccactttcca agggagggcg tgttcgtgtc caacggcacc 3300
cactggtttg tgacacagcg caatttctac gagccccaga tcatcaccac agacaacacc 3360
ttcgtgagcg gcaactgtga cgtggtcatc ggcatcgtga acaataccgt gtatgatcca 3420
ctccagcccg agctggacag ctttaaggag gagctggata agtatttcaa gaatcacacc 3480
tcccctgacg tggatctggg cgacatcagc ggcatcaatg cctccgtggt gaacatccag 3540
aaggagatcg accgcctgaa cgaggtggct aagaatctga acgagagcct gatcgacctc 3600
caggagctgg gcaagtatga gcagtacatc aagtggccct ggtacatctg gctgggcttc 3660
atcgccggcc tgatcgccat cgtgatggtg accatcatgc tgtgctgtat gacatcctgc 3720
tgttcttgcc tgaagggctg ctgtagctgt ggctcctgct gtaagtttga cgaggatgac 3780
tctgaacctg tgctgaaggg cgtgaagctg cattacacct aa 3822
<210> 18
<211> 1273
<212> PRT
<213> SARS-CoV-2 (Wuhan strain S-2P strain)
<400> 18
Met Phe Val Phe Leu Val Leu Leu Pro Leu Val Ser Ser Gln Cys Val
1 5 10 15
Asn Leu Thr Thr Arg Thr Gln Leu Pro Pro Ala Tyr Thr Asn Ser Phe
20 25 30
Thr Arg Gly Val Tyr Tyr Pro Asp Lys Val Phe Arg Ser Ser Val Leu
35 40 45
His Ser Thr Gln Asp Leu Phe Leu Pro Phe Phe Ser Asn Val Thr Trp
50 55 60
Phe His Ala Ile His Val Ser Gly Thr Asn Gly Thr Lys Arg Phe Asp
65 70 75 80
Asn Pro Val Leu Pro Phe Asn Asp Gly Val Tyr Phe Ala Ser Thr Glu
85 90 95
Lys Ser Asn Ile Ile Arg Gly Trp Ile Phe Gly Thr Thr Leu Asp Ser
100 105 110
Lys Thr Gln Ser Leu Leu Ile Val Asn Asn Ala Thr Asn Val Val Ile
115 120 125
Lys Val Cys Glu Phe Gln Phe Cys Asn Asp Pro Phe Leu Gly Val Tyr
130 135 140
Tyr His Lys Asn Asn Lys Ser Trp Met Glu Ser Glu Phe Arg Val Tyr
145 150 155 160
Ser Ser Ala Asn Asn Cys Thr Phe Glu Tyr Val Ser Gln Pro Phe Leu
165 170 175
Met Asp Leu Glu Gly Lys Gln Gly Asn Phe Lys Asn Leu Arg Glu Phe
180 185 190
Val Phe Lys Asn Ile Asp Gly Tyr Phe Lys Ile Tyr Ser Lys His Thr
195 200 205
Pro Ile Asn Leu Val Arg Asp Leu Pro Gln Gly Phe Ser Ala Leu Glu
210 215 220
Pro Leu Val Asp Leu Pro Ile Gly Ile Asn Ile Thr Arg Phe Gln Thr
225 230 235 240
Leu Leu Ala Leu His Arg Ser Tyr Leu Thr Pro Gly Asp Ser Ser Ser
245 250 255
Gly Trp Thr Ala Gly Ala Ala Ala Tyr Tyr Val Gly Tyr Leu Gln Pro
260 265 270
Arg Thr Phe Leu Leu Lys Tyr Asn Glu Asn Gly Thr Ile Thr Asp Ala
275 280 285
Val Asp Cys Ala Leu Asp Pro Leu Ser Glu Thr Lys Cys Thr Leu Lys
290 295 300
Ser Phe Thr Val Glu Lys Gly Ile Tyr Gln Thr Ser Asn Phe Arg Val
305 310 315 320
Gln Pro Thr Glu Ser Ile Val Arg Phe Pro Asn Ile Thr Asn Leu Cys
325 330 335
Pro Phe Gly Glu Val Phe Asn Ala Thr Arg Phe Ala Ser Val Tyr Ala
340 345 350
Trp Asn Arg Lys Arg Ile Ser Asn Cys Val Ala Asp Tyr Ser Val Leu
355 360 365
Tyr Asn Ser Ala Ser Phe Ser Thr Phe Lys Cys Tyr Gly Val Ser Pro
370 375 380
Thr Lys Leu Asn Asp Leu Cys Phe Thr Asn Val Tyr Ala Asp Ser Phe
385 390 395 400
Val Ile Arg Gly Asp Glu Val Arg Gln Ile Ala Pro Gly Gln Thr Gly
405 410 415
Lys Ile Ala Asp Tyr Asn Tyr Lys Leu Pro Asp Asp Phe Thr Gly Cys
420 425 430
Val Ile Ala Trp Asn Ser Asn Asn Leu Asp Ser Lys Val Gly Gly Asn
435 440 445
Tyr Asn Tyr Leu Tyr Arg Leu Phe Arg Lys Ser Asn Leu Lys Pro Phe
450 455 460
Glu Arg Asp Ile Ser Thr Glu Ile Tyr Gln Ala Gly Ser Thr Pro Cys
465 470 475 480
Asn Gly Val Glu Gly Phe Asn Cys Tyr Phe Pro Leu Gln Ser Tyr Gly
485 490 495
Phe Gln Pro Thr Asn Gly Val Gly Tyr Gln Pro Tyr Arg Val Val Val
500 505 510
Leu Ser Phe Glu Leu Leu His Ala Pro Ala Thr Val Cys Gly Pro Lys
515 520 525
Lys Ser Thr Asn Leu Val Lys Asn Lys Cys Val Asn Phe Asn Phe Asn
530 535 540
Gly Leu Thr Gly Thr Gly Val Leu Thr Glu Ser Asn Lys Lys Phe Leu
545 550 555 560
Pro Phe Gln Gln Phe Gly Arg Asp Ile Ala Asp Thr Thr Asp Ala Val
565 570 575
Arg Asp Pro Gln Thr Leu Glu Ile Leu Asp Ile Thr Pro Cys Ser Phe
580 585 590
Gly Gly Val Ser Val Ile Thr Pro Gly Thr Asn Thr Ser Asn Gln Val
595 600 605
Ala Val Leu Tyr Gln Asp Val Asn Cys Thr Glu Val Pro Val Ala Ile
610 615 620
His Ala Asp Gln Leu Thr Pro Thr Trp Arg Val Tyr Ser Thr Gly Ser
625 630 635 640
Asn Val Phe Gln Thr Arg Ala Gly Cys Leu Ile Gly Ala Glu His Val
645 650 655
Asn Asn Ser Tyr Glu Cys Asp Ile Pro Ile Gly Ala Gly Ile Cys Ala
660 665 670
Ser Tyr Gln Thr Gln Thr Asn Ser Pro Arg Arg Ala Arg Ser Val Ala
675 680 685
Ser Gln Ser Ile Ile Ala Tyr Thr Met Ser Leu Gly Ala Glu Asn Ser
690 695 700
Val Ala Tyr Ser Asn Asn Ser Ile Ala Ile Pro Thr Asn Phe Thr Ile
705 710 715 720
Ser Val Thr Thr Glu Ile Leu Pro Val Ser Met Thr Lys Thr Ser Val
725 730 735
Asp Cys Thr Met Tyr Ile Cys Gly Asp Ser Thr Glu Cys Ser Asn Leu
740 745 750
Leu Leu Gln Tyr Gly Ser Phe Cys Thr Gln Leu Asn Arg Ala Leu Thr
755 760 765
Gly Ile Ala Val Glu Gln Asp Lys Asn Thr Gln Glu Val Phe Ala Gln
770 775 780
Val Lys Gln Ile Tyr Lys Thr Pro Pro Ile Lys Asp Phe Gly Gly Phe
785 790 795 800
Asn Phe Ser Gln Ile Leu Pro Asp Pro Ser Lys Pro Ser Lys Arg Ser
805 810 815
Phe Ile Glu Asp Leu Leu Phe Asn Lys Val Thr Leu Ala Asp Ala Gly
820 825 830
Phe Ile Lys Gln Tyr Gly Asp Cys Leu Gly Asp Ile Ala Ala Arg Asp
835 840 845
Leu Ile Cys Ala Gln Lys Phe Asn Gly Leu Thr Val Leu Pro Pro Leu
850 855 860
Leu Thr Asp Glu Met Ile Ala Gln Tyr Thr Ser Ala Leu Leu Ala Gly
865 870 875 880
Thr Ile Thr Ser Gly Trp Thr Phe Gly Ala Gly Ala Ala Leu Gln Ile
885 890 895
Pro Phe Ala Met Gln Met Ala Tyr Arg Phe Asn Gly Ile Gly Val Thr
900 905 910
Gln Asn Val Leu Tyr Glu Asn Gln Lys Leu Ile Ala Asn Gln Phe Asn
915 920 925
Ser Ala Ile Gly Lys Ile Gln Asp Ser Leu Ser Ser Thr Ala Ser Ala
930 935 940
Leu Gly Lys Leu Gln Asp Val Val Asn Gln Asn Ala Gln Ala Leu Asn
945 950 955 960
Thr Leu Val Lys Gln Leu Ser Ser Asn Phe Gly Ala Ile Ser Ser Val
965 970 975
Leu Asn Asp Ile Leu Ser Arg Leu Asp Pro Pro Glu Ala Glu Val Gln
980 985 990
Ile Asp Arg Leu Ile Thr Gly Arg Leu Gln Ser Leu Gln Thr Tyr Val
995 1000 1005
Thr Gln Gln Leu Ile Arg Ala Ala Glu Ile Arg Ala Ser Ala Asn
1010 1015 1020
Leu Ala Ala Thr Lys Met Ser Glu Cys Val Leu Gly Gln Ser Lys
1025 1030 1035
Arg Val Asp Phe Cys Gly Lys Gly Tyr His Leu Met Ser Phe Pro
1040 1045 1050
Gln Ser Ala Pro His Gly Val Val Phe Leu His Val Thr Tyr Val
1055 1060 1065
Pro Ala Gln Glu Lys Asn Phe Thr Thr Ala Pro Ala Ile Cys His
1070 1075 1080
Asp Gly Lys Ala His Phe Pro Arg Glu Gly Val Phe Val Ser Asn
1085 1090 1095
Gly Thr His Trp Phe Val Thr Gln Arg Asn Phe Tyr Glu Pro Gln
1100 1105 1110
Ile Ile Thr Thr Asp Asn Thr Phe Val Ser Gly Asn Cys Asp Val
1115 1120 1125
Val Ile Gly Ile Val Asn Asn Thr Val Tyr Asp Pro Leu Gln Pro
1130 1135 1140
Glu Leu Asp Ser Phe Lys Glu Glu Leu Asp Lys Tyr Phe Lys Asn
1145 1150 1155
His Thr Ser Pro Asp Val Asp Leu Gly Asp Ile Ser Gly Ile Asn
1160 1165 1170
Ala Ser Val Val Asn Ile Gln Lys Glu Ile Asp Arg Leu Asn Glu
1175 1180 1185
Val Ala Lys Asn Leu Asn Glu Ser Leu Ile Asp Leu Gln Glu Leu
1190 1195 1200
Gly Lys Tyr Glu Gln Tyr Ile Lys Trp Pro Trp Tyr Ile Trp Leu
1205 1210 1215
Gly Phe Ile Ala Gly Leu Ile Ala Ile Val Met Val Thr Ile Met
1220 1225 1230
Leu Cys Cys Met Thr Ser Cys Cys Ser Cys Leu Lys Gly Cys Cys
1235 1240 1245
Ser Cys Gly Ser Cys Cys Lys Phe Asp Glu Asp Asp Ser Glu Pro
1250 1255 1260
Val Leu Lys Gly Val Lys Leu His Tyr Thr
1265 1270
<210> 19
<211> 3816
<212> DNA
<213> SARS-CoV-2 (Delta S-2P strain)
<400> 19
atgttcgtct tcctggtcct gctgcctctg gtctcctcac agtgcgtcaa tctgcgaact 60
cggactcagc tgccacctgc ttatactaat agcttcacca gaggcgtgta ctatcctgac 120
aaggtgttta gaagctccgt gctgcactct acacaggatc tgtttctgcc attctttagc 180
aacgtgacct ggttccacgc catccacgtg agcggcacca atggcacaaa gcggttcgac 240
aatcccgtgc tgccttttaa cgatggcgtg tacttcgcct ctatcgagaa gagcaacatc 300
atcagaggct ggatctttgg caccacactg gactccaaga cacagtctct gctgatcgtg 360
aacaatgcca ccaacgtggt catcaaggtg tgcgagttcc agttttgtaa tgatcccttc 420
ctggacgtgt actatcacaa gaacaataag agctggatgg agtccggagt gtattctagc 480
gccaacaact gcacatttga gtacgtgagc cagcctttcc tgatggacct ggagggcaag 540
cagggcaatt tcaagaacct gagggagttc gtgtttaaga atatcgacgg ctacttcaaa 600
atctactcta agcacacccc catcaacctg gtgcgcgacc tgcctcaggg cttcagcgcc 660
ctggagcccc tggtggatct gcctatcggc atcaacatca cccggtttca gacactgctg 720
gccctgcaca gaagctacct gacacccggc gactcctcta gcggatggac cgccggcgct 780
gccgcctact atgtgggcta cctccagccc cggaccttcc tgctgaagta caacgagaat 840
ggcaccatca cagacgcagt ggattgcgcc ctggaccccc tgagcgagac aaagtgtaca 900
ctgaagtcct ttaccgtgga gaagggcatc tatcagacat ccaatttcag ggtgcagcca 960
accgagtcta tcgtgcgctt tcctaatatc acaaacctgt gcccatttgg cgaggtgttc 1020
aacgcaaccc gcttcgccag cgtgtacgcc tggaatagga agcggatcag caactgcgtg 1080
gccgactata gcgtgctgta caactccgcc tctttcagca cctttaagtg ctatggcgtg 1140
tcccccacaa agctgaatga cctgtgcttt accaacgtct acgccgattc tttcgtgatc 1200
aggggcgacg aggtgcgcca gatcgccccc ggccagacag gcaagatcgc agactacaat 1260
tataagctgc cagacgattt caccggctgc gtgatcgcct ggaacagcaa caatctggat 1320
tccaaagtgg gcggcaacta caattatcgg taccggctgt ttagaaagag caatctgaag 1380
cccttcgaga gggacatctc tacagaaatc taccaggccg gcagcaagcc ttgcaatggc 1440
gtggagggct ttaactgtta tttcccactc cagtcctacg gcttccagcc cacaaacggc 1500
gtgggctatc agccttaccg cgtggtggtg ctgagctttg agctgctgca cgccccagca 1560
acagtgtgcg gccccaagaa gtccaccaat ctggtgaaga acaagtgcgt gaacttcaac 1620
ttcaacggcc tgaccggcac aggcgtgctg accgagtcca acaagaagtt cctgccattt 1680
cagcagttcg gcagggacat cgcagatacc acagacgccg tgcgcgaccc acagaccctg 1740
gagatcctgg acatcacacc ctgctctttc ggcggcgtga gcgtgatcac acccggcacc 1800
aatacaagca accaggtggc cgtgctgtat cagggcgtga attgtaccga ggtgcccgtg 1860
gctatccacg ccgatcagct gaccccaaca tggcgggtgt acagcaccgg ctccaacgtc 1920
ttccagacaa gagccggatg cctgatcgga gcagagcacg tgaacaattc ctatgagtgc 1980
gacatcccaa tcggcgccgg catctgtgcc tcttaccaga cccagacaaa ctctcgcaga 2040
agagcccgga gcgtggcctc ccagtctatc atcgcctata ccatgtccct gggcgccgag 2100
aacagcgtgg cctactctaa caatagcatc gccatcccaa ccaacttcac aatctctgtg 2160
accacagaga tcctgcccgt gtccatgacc aagacatctg tggactgcac aatgtatatc 2220
tgtggcgatt ctaccgagtg cagcaacctg ctgctccagt acggcagctt ttgtacccag 2280
ctgaatagag ccctgacagg catcgccgtg gagcaggata agaacacaca ggaggtgttc 2340
gcccaggtga agcaaatcta caagaccccc cctatcaagg actttggcgg cttcaatttt 2400
tcccagatcc tgcctgatcc atccaagcct tctaagcgga gctttatcga ggacctgctg 2460
ttcaacaagg tgaccctggc cgatgccggc ttcatcaagc agtatggcga ttgcctgggc 2520
gacatcgcag ccagggacct gatctgcgcc cagaagttta atggcctgac cgtgctgcca 2580
cccctgctga cagatgagat gatcgcacag tacacaagcg ccctgctggc cggcaccatc 2640
acatccggat ggaccttcgg cgcaggagcc gccctccaga tcccctttgc catgcagatg 2700
gcctataggt tcaacggcat cggcgtgacc cagaatgtgc tgtacgagaa ccagaagctg 2760
atcgccaatc agtttaactc cgccatcggc aagatccagg acagcctgtc ctctacagcc 2820
agcgccctgg gcaagctcca gaatgtggtg aatcagaacg cccaggccct gaataccctg 2880
gtgaagcagc tgagcagcaa cttcggcgcc atctctagcg tgctgaatga catcctgagc 2940
cggctggacc cgccggaggc agaggtgcag atcgaccggc tgatcaccgg ccggctccag 3000
agcctccaga cctatgtgac acagcagctg atcagggccg ccgagatcag ggccagcgcc 3060
aatctggcag caaccaagat gtccgagtgc gtgctgggcc agtctaagag agtggacttt 3120
tgtggcaagg gctatcacct gatgtccttc cctcagtctg ccccacacgg cgtggtgttt 3180
ctgcacgtga cctacgtgcc cgcccaggag aagaacttca ccacagcccc tgccatctgc 3240
cacgatggca aggcccactt tccaagggag ggcgtgttcg tgtccaacgg cacccactgg 3300
tttgtgacac agcgcaattt ctacgagccc cagatcatca ccacagacaa caccttcgtg 3360
agcggcaact gtgacgtggt catcggcatc gtgaacaata ccgtgtatga tccactccag 3420
cccgagctgg acagctttaa ggaggagctg gataagtatt tcaagaatca cacctcccct 3480
gacgtggatc tgggcgacat cagcggcatc aatgcctccg tggtgaacat ccagaaggag 3540
atcgaccgcc tgaacgaggt ggctaagaat ctgaacgaga gcctgatcga cctccaggag 3600
ctgggcaagt atgagcagta catcaagtgg ccctggtaca tctggctggg cttcatcgcc 3660
ggcctgatcg ccatcgtgat ggtgaccatc atgctgtgct gtatgacatc ctgctgttct 3720
tgcctgaagg gctgctgtag ctgtggctcc tgctgtaagt ttgacgagga tgactctgaa 3780
cctgtgctga agggcgtgaa gctgcattac acctaa 3816
<210> 20
<211> 1271
<212> PRT
<213> SARS-CoV-2 (Delta S-2P strain)
<400> 20
Met Phe Val Phe Leu Val Leu Leu Pro Leu Val Ser Ser Gln Cys Val
1 5 10 15
Asn Leu Arg Thr Arg Thr Gln Leu Pro Pro Ala Tyr Thr Asn Ser Phe
20 25 30
Thr Arg Gly Val Tyr Tyr Pro Asp Lys Val Phe Arg Ser Ser Val Leu
35 40 45
His Ser Thr Gln Asp Leu Phe Leu Pro Phe Phe Ser Asn Val Thr Trp
50 55 60
Phe His Ala Ile His Val Ser Gly Thr Asn Gly Thr Lys Arg Phe Asp
65 70 75 80
Asn Pro Val Leu Pro Phe Asn Asp Gly Val Tyr Phe Ala Ser Ile Glu
85 90 95
Lys Ser Asn Ile Ile Arg Gly Trp Ile Phe Gly Thr Thr Leu Asp Ser
100 105 110
Lys Thr Gln Ser Leu Leu Ile Val Asn Asn Ala Thr Asn Val Val Ile
115 120 125
Lys Val Cys Glu Phe Gln Phe Cys Asn Asp Pro Phe Leu Asp Val Tyr
130 135 140
Tyr His Lys Asn Asn Lys Ser Trp Met Glu Ser Gly Val Tyr Ser Ser
145 150 155 160
Ala Asn Asn Cys Thr Phe Glu Tyr Val Ser Gln Pro Phe Leu Met Asp
165 170 175
Leu Glu Gly Lys Gln Gly Asn Phe Lys Asn Leu Arg Glu Phe Val Phe
180 185 190
Lys Asn Ile Asp Gly Tyr Phe Lys Ile Tyr Ser Lys His Thr Pro Ile
195 200 205
Asn Leu Val Arg Asp Leu Pro Gln Gly Phe Ser Ala Leu Glu Pro Leu
210 215 220
Val Asp Leu Pro Ile Gly Ile Asn Ile Thr Arg Phe Gln Thr Leu Leu
225 230 235 240
Ala Leu His Arg Ser Tyr Leu Thr Pro Gly Asp Ser Ser Ser Gly Trp
245 250 255
Thr Ala Gly Ala Ala Ala Tyr Tyr Val Gly Tyr Leu Gln Pro Arg Thr
260 265 270
Phe Leu Leu Lys Tyr Asn Glu Asn Gly Thr Ile Thr Asp Ala Val Asp
275 280 285
Cys Ala Leu Asp Pro Leu Ser Glu Thr Lys Cys Thr Leu Lys Ser Phe
290 295 300
Thr Val Glu Lys Gly Ile Tyr Gln Thr Ser Asn Phe Arg Val Gln Pro
305 310 315 320
Thr Glu Ser Ile Val Arg Phe Pro Asn Ile Thr Asn Leu Cys Pro Phe
325 330 335
Gly Glu Val Phe Asn Ala Thr Arg Phe Ala Ser Val Tyr Ala Trp Asn
340 345 350
Arg Lys Arg Ile Ser Asn Cys Val Ala Asp Tyr Ser Val Leu Tyr Asn
355 360 365
Ser Ala Ser Phe Ser Thr Phe Lys Cys Tyr Gly Val Ser Pro Thr Lys
370 375 380
Leu Asn Asp Leu Cys Phe Thr Asn Val Tyr Ala Asp Ser Phe Val Ile
385 390 395 400
Arg Gly Asp Glu Val Arg Gln Ile Ala Pro Gly Gln Thr Gly Lys Ile
405 410 415
Ala Asp Tyr Asn Tyr Lys Leu Pro Asp Asp Phe Thr Gly Cys Val Ile
420 425 430
Ala Trp Asn Ser Asn Asn Leu Asp Ser Lys Val Gly Gly Asn Tyr Asn
435 440 445
Tyr Arg Tyr Arg Leu Phe Arg Lys Ser Asn Leu Lys Pro Phe Glu Arg
450 455 460
Asp Ile Ser Thr Glu Ile Tyr Gln Ala Gly Ser Lys Pro Cys Asn Gly
465 470 475 480
Val Glu Gly Phe Asn Cys Tyr Phe Pro Leu Gln Ser Tyr Gly Phe Gln
485 490 495
Pro Thr Asn Gly Val Gly Tyr Gln Pro Tyr Arg Val Val Val Leu Ser
500 505 510
Phe Glu Leu Leu His Ala Pro Ala Thr Val Cys Gly Pro Lys Lys Ser
515 520 525
Thr Asn Leu Val Lys Asn Lys Cys Val Asn Phe Asn Phe Asn Gly Leu
530 535 540
Thr Gly Thr Gly Val Leu Thr Glu Ser Asn Lys Lys Phe Leu Pro Phe
545 550 555 560
Gln Gln Phe Gly Arg Asp Ile Ala Asp Thr Thr Asp Ala Val Arg Asp
565 570 575
Pro Gln Thr Leu Glu Ile Leu Asp Ile Thr Pro Cys Ser Phe Gly Gly
580 585 590
Val Ser Val Ile Thr Pro Gly Thr Asn Thr Ser Asn Gln Val Ala Val
595 600 605
Leu Tyr Gln Gly Val Asn Cys Thr Glu Val Pro Val Ala Ile His Ala
610 615 620
Asp Gln Leu Thr Pro Thr Trp Arg Val Tyr Ser Thr Gly Ser Asn Val
625 630 635 640
Phe Gln Thr Arg Ala Gly Cys Leu Ile Gly Ala Glu His Val Asn Asn
645 650 655
Ser Tyr Glu Cys Asp Ile Pro Ile Gly Ala Gly Ile Cys Ala Ser Tyr
660 665 670
Gln Thr Gln Thr Asn Ser Arg Arg Arg Ala Arg Ser Val Ala Ser Gln
675 680 685
Ser Ile Ile Ala Tyr Thr Met Ser Leu Gly Ala Glu Asn Ser Val Ala
690 695 700
Tyr Ser Asn Asn Ser Ile Ala Ile Pro Thr Asn Phe Thr Ile Ser Val
705 710 715 720
Thr Thr Glu Ile Leu Pro Val Ser Met Thr Lys Thr Ser Val Asp Cys
725 730 735
Thr Met Tyr Ile Cys Gly Asp Ser Thr Glu Cys Ser Asn Leu Leu Leu
740 745 750
Gln Tyr Gly Ser Phe Cys Thr Gln Leu Asn Arg Ala Leu Thr Gly Ile
755 760 765
Ala Val Glu Gln Asp Lys Asn Thr Gln Glu Val Phe Ala Gln Val Lys
770 775 780
Gln Ile Tyr Lys Thr Pro Pro Ile Lys Asp Phe Gly Gly Phe Asn Phe
785 790 795 800
Ser Gln Ile Leu Pro Asp Pro Ser Lys Pro Ser Lys Arg Ser Phe Ile
805 810 815
Glu Asp Leu Leu Phe Asn Lys Val Thr Leu Ala Asp Ala Gly Phe Ile
820 825 830
Lys Gln Tyr Gly Asp Cys Leu Gly Asp Ile Ala Ala Arg Asp Leu Ile
835 840 845
Cys Ala Gln Lys Phe Asn Gly Leu Thr Val Leu Pro Pro Leu Leu Thr
850 855 860
Asp Glu Met Ile Ala Gln Tyr Thr Ser Ala Leu Leu Ala Gly Thr Ile
865 870 875 880
Thr Ser Gly Trp Thr Phe Gly Ala Gly Ala Ala Leu Gln Ile Pro Phe
885 890 895
Ala Met Gln Met Ala Tyr Arg Phe Asn Gly Ile Gly Val Thr Gln Asn
900 905 910
Val Leu Tyr Glu Asn Gln Lys Leu Ile Ala Asn Gln Phe Asn Ser Ala
915 920 925
Ile Gly Lys Ile Gln Asp Ser Leu Ser Ser Thr Ala Ser Ala Leu Gly
930 935 940
Lys Leu Gln Asn Val Val Asn Gln Asn Ala Gln Ala Leu Asn Thr Leu
945 950 955 960
Val Lys Gln Leu Ser Ser Asn Phe Gly Ala Ile Ser Ser Val Leu Asn
965 970 975
Asp Ile Leu Ser Arg Leu Asp Pro Pro Glu Ala Glu Val Gln Ile Asp
980 985 990
Arg Leu Ile Thr Gly Arg Leu Gln Ser Leu Gln Thr Tyr Val Thr Gln
995 1000 1005
Gln Leu Ile Arg Ala Ala Glu Ile Arg Ala Ser Ala Asn Leu Ala
1010 1015 1020
Ala Thr Lys Met Ser Glu Cys Val Leu Gly Gln Ser Lys Arg Val
1025 1030 1035
Asp Phe Cys Gly Lys Gly Tyr His Leu Met Ser Phe Pro Gln Ser
1040 1045 1050
Ala Pro His Gly Val Val Phe Leu His Val Thr Tyr Val Pro Ala
1055 1060 1065
Gln Glu Lys Asn Phe Thr Thr Ala Pro Ala Ile Cys His Asp Gly
1070 1075 1080
Lys Ala His Phe Pro Arg Glu Gly Val Phe Val Ser Asn Gly Thr
1085 1090 1095
His Trp Phe Val Thr Gln Arg Asn Phe Tyr Glu Pro Gln Ile Ile
1100 1105 1110
Thr Thr Asp Asn Thr Phe Val Ser Gly Asn Cys Asp Val Val Ile
1115 1120 1125
Gly Ile Val Asn Asn Thr Val Tyr Asp Pro Leu Gln Pro Glu Leu
1130 1135 1140
Asp Ser Phe Lys Glu Glu Leu Asp Lys Tyr Phe Lys Asn His Thr
1145 1150 1155
Ser Pro Asp Val Asp Leu Gly Asp Ile Ser Gly Ile Asn Ala Ser
1160 1165 1170
Val Val Asn Ile Gln Lys Glu Ile Asp Arg Leu Asn Glu Val Ala
1175 1180 1185
Lys Asn Leu Asn Glu Ser Leu Ile Asp Leu Gln Glu Leu Gly Lys
1190 1195 1200
Tyr Glu Gln Tyr Ile Lys Trp Pro Trp Tyr Ile Trp Leu Gly Phe
1205 1210 1215
Ile Ala Gly Leu Ile Ala Ile Val Met Val Thr Ile Met Leu Cys
1220 1225 1230
Cys Met Thr Ser Cys Cys Ser Cys Leu Lys Gly Cys Cys Ser Cys
1235 1240 1245
Gly Ser Cys Cys Lys Phe Asp Glu Asp Asp Ser Glu Pro Val Leu
1250 1255 1260
Lys Gly Val Lys Leu His Tyr Thr
1265 1270
<210> 21
<211> 3816
<212> DNA
<213> Artificial Sequence
<220>
<223> S-(deg-RBD) for Delta strain
<400> 21
atgttcgtct tcctggtcct gctgcctctg gtctcctcac agtgcgtcaa tctgcgaact 60
cggactcagc tgccacctgc ttatactaat agcttcacca gaggcgtgta ctatcctgac 120
aaggtgttta gaagctccgt gctgcactct acacaggatc tgtttctgcc attctttagc 180
aacgtgacct ggttccacgc catccacgtg agcggcacca atggcacaaa gcggttcgac 240
aatcccgtgc tgccttttaa cgatggcgtg tacttcgcct ctatcgagaa gagcaacatc 300
atcagaggct ggatctttgg caccacactg gactccaaga cacagtctct gctgatcgtg 360
aacaatgcca ccaacgtggt catcaaggtg tgcgagttcc agttttgtaa tgatcccttc 420
ctggacgtgt actatcacaa gaacaataag agctggatgg agtccggagt gtattctagc 480
gccaacaact gcacatttga gtacgtgagc cagcctttcc tgatggacct ggagggcaag 540
cagggcaatt tcaagaacct gagggagttc gtgtttaaga atatcgacgg ctacttcaaa 600
atctactcta agcacacccc catcaacctg gtgcgcgacc tgcctcaggg cttcagcgcc 660
ctggagcccc tggtggatct gcctatcggc atcaacatca cccggtttca gacactgctg 720
gccctgcaca gaagctacct gacacccggc gactcctcta gcggatggac cgccggcgct 780
gccgcctact atgtgggcta cctccagccc cggaccttcc tgctgaagta caacgagaat 840
ggcaccatca cagacgcagt ggattgcgcc ctggaccccc tgagcgagac aaagtgtaca 900
ctgaagtcct ttaccgtgga gaagggcatc tatcagacat ccaatttcag ggtgcagcca 960
gccgaggcga tcgtgcgctt tcctgaaatc acaaacctgt gcccatttgg cgaggtgttc 1020
gaggcaaccc gcttcgccag cgtgtacgcc tggaatagga agcggatcag caactgcgtg 1080
gccgactata gcgtgctgta caactccgcc tctttcagca cctttaagtg ctatggcgtg 1140
tcccccacaa agctgaatga cctgtgcttt accaacgtct acgccgattc tttcgtgatc 1200
aggggcgacg aggtgcgcca gatcgccccc ggccagacag gcaagatcgc agactacaat 1260
tataagctgc cagacgattt caccggctgc gtgatcgcct ggaacagcaa caatctggat 1320
tccaaagtgg gcggcaacta caattatcgg taccggctgt ttagaaagag caatctgaag 1380
cccttcgaga gggacatctc tacagaaatc taccaggccg gcagcaagcc ttgcaatggc 1440
gtggagggct ttaactgtta tttcccactc cagtcctacg gcttccagcc cacaaacggc 1500
gtgggctatc agccttaccg cgtggtggtg ctgagctttg agctgctgca cgccccagca 1560
acagtgtgcg gccccaagaa gtccaccaat ctggtgaaga acaagtgcgt gaacttcaac 1620
ttcaacggcc tgaccggcac aggcgtgctg accgagtcca acaagaagtt cctgccattt 1680
cagcagttcg gcagggacat cgcagatacc acagacgccg tgcgcgaccc acagaccctg 1740
gagatcctgg acatcacacc ctgctctttc ggcggcgtga gcgtgatcac acccggcacc 1800
aatacaagca accaggtggc cgtgctgtat cagggcgtga attgtaccga ggtgcccgtg 1860
gctatccacg ccgatcagct gaccccaaca tggcgggtgt acagcaccgg ctccaacgtc 1920
ttccagacaa gagccggatg cctgatcgga gcagagcacg tgaacaattc ctatgagtgc 1980
gacatcccaa tcggcgccgg catctgtgcc tcttaccaga cccagacaaa ctctcgcaga 2040
agagcccgga gcgtggcctc ccagtctatc atcgcctata ccatgtccct gggcgccgag 2100
aacagcgtgg cctactctaa caatagcatc gccatcccaa ccaacttcac aatctctgtg 2160
accacagaga tcctgcccgt gtccatgacc aagacatctg tggactgcac aatgtatatc 2220
tgtggcgatt ctaccgagtg cagcaacctg ctgctccagt acggcagctt ttgtacccag 2280
ctgaatagag ccctgacagg catcgccgtg gagcaggata agaacacaca ggaggtgttc 2340
gcccaggtga agcaaatcta caagaccccc cctatcaagg actttggcgg cttcaatttt 2400
tcccagatcc tgcctgatcc atccaagcct tctaagcgga gctttatcga ggacctgctg 2460
ttcaacaagg tgaccctggc cgatgccggc ttcatcaagc agtatggcga ttgcctgggc 2520
gacatcgcag ccagggacct gatctgcgcc cagaagttta atggcctgac cgtgctgcca 2580
cccctgctga cagatgagat gatcgcacag tacacaagcg ccctgctggc cggcaccatc 2640
acatccggat ggaccttcgg cgcaggagcc gccctccaga tcccctttgc catgcagatg 2700
gcctataggt tcaacggcat cggcgtgacc cagaatgtgc tgtacgagaa ccagaagctg 2760
atcgccaatc agtttaactc cgccatcggc aagatccagg acagcctgtc ctctacagcc 2820
agcgccctgg gcaagctcca gaatgtggtg aatcagaacg cccaggccct gaataccctg 2880
gtgaagcagc tgagcagcaa cttcggcgcc atctctagcg tgctgaatga catcctgagc 2940
cggctggaca aggtggaggc agaggtgcag atcgaccggc tgatcaccgg ccggctccag 3000
agcctccaga cctatgtgac acagcagctg atcagggccg ccgagatcag ggccagcgcc 3060
aatctggcag caaccaagat gtccgagtgc gtgctgggcc agtctaagag agtggacttt 3120
tgtggcaagg gctatcacct gatgtccttc cctcagtctg ccccacacgg cgtggtgttt 3180
ctgcacgtga cctacgtgcc cgcccaggag aagaacttca ccacagcccc tgccatctgc 3240
cacgatggca aggcccactt tccaagggag ggcgtgttcg tgtccaacgg cacccactgg 3300
tttgtgacac agcgcaattt ctacgagccc cagatcatca ccacagacaa caccttcgtg 3360
agcggcaact gtgacgtggt catcggcatc gtgaacaata ccgtgtatga tccactccag 3420
cccgagctgg acagctttaa ggaggagctg gataagtatt tcaagaatca cacctcccct 3480
gacgtggatc tgggcgacat cagcggcatc aatgcctccg tggtgaacat ccagaaggag 3540
atcgaccgcc tgaacgaggt ggctaagaat ctgaacgaga gcctgatcga cctccaggag 3600
ctgggcaagt atgagcagta catcaagtgg ccctggtaca tctggctggg cttcatcgcc 3660
ggcctgatcg ccatcgtgat ggtgaccatc atgctgtgct gtatgacatc ctgctgttct 3720
tgcctgaagg gctgctgtag ctgtggctcc tgctgtaagt ttgacgagga tgactctgaa 3780
cctgtgctga agggcgtgaa gctgcattac acctaa 3816
<210> 22
<211> 1271
<212> PRT
<213> Artificial Sequence
<220>
<223> S-(deg-RBD) for Delta strain
<400> 22
Met Phe Val Phe Leu Val Leu Leu Pro Leu Val Ser Ser Gln Cys Val
1 5 10 15
Asn Leu Arg Thr Arg Thr Gln Leu Pro Pro Ala Tyr Thr Asn Ser Phe
20 25 30
Thr Arg Gly Val Tyr Tyr Pro Asp Lys Val Phe Arg Ser Ser Val Leu
35 40 45
His Ser Thr Gln Asp Leu Phe Leu Pro Phe Phe Ser Asn Val Thr Trp
50 55 60
Phe His Ala Ile His Val Ser Gly Thr Asn Gly Thr Lys Arg Phe Asp
65 70 75 80
Asn Pro Val Leu Pro Phe Asn Asp Gly Val Tyr Phe Ala Ser Ile Glu
85 90 95
Lys Ser Asn Ile Ile Arg Gly Trp Ile Phe Gly Thr Thr Leu Asp Ser
100 105 110
Lys Thr Gln Ser Leu Leu Ile Val Asn Asn Ala Thr Asn Val Val Ile
115 120 125
Lys Val Cys Glu Phe Gln Phe Cys Asn Asp Pro Phe Leu Asp Val Tyr
130 135 140
Tyr His Lys Asn Asn Lys Ser Trp Met Glu Ser Gly Val Tyr Ser Ser
145 150 155 160
Ala Asn Asn Cys Thr Phe Glu Tyr Val Ser Gln Pro Phe Leu Met Asp
165 170 175
Leu Glu Gly Lys Gln Gly Asn Phe Lys Asn Leu Arg Glu Phe Val Phe
180 185 190
Lys Asn Ile Asp Gly Tyr Phe Lys Ile Tyr Ser Lys His Thr Pro Ile
195 200 205
Asn Leu Val Arg Asp Leu Pro Gln Gly Phe Ser Ala Leu Glu Pro Leu
210 215 220
Val Asp Leu Pro Ile Gly Ile Asn Ile Thr Arg Phe Gln Thr Leu Leu
225 230 235 240
Ala Leu His Arg Ser Tyr Leu Thr Pro Gly Asp Ser Ser Ser Gly Trp
245 250 255
Thr Ala Gly Ala Ala Ala Tyr Tyr Val Gly Tyr Leu Gln Pro Arg Thr
260 265 270
Phe Leu Leu Lys Tyr Asn Glu Asn Gly Thr Ile Thr Asp Ala Val Asp
275 280 285
Cys Ala Leu Asp Pro Leu Ser Glu Thr Lys Cys Thr Leu Lys Ser Phe
290 295 300
Thr Val Glu Lys Gly Ile Tyr Gln Thr Ser Asn Phe Arg Val Gln Pro
305 310 315 320
Ala Glu Ala Ile Val Arg Phe Pro Gln Ile Thr Asn Leu Cys Pro Phe
325 330 335
Gly Glu Val Phe Gln Ala Thr Arg Phe Ala Ser Val Tyr Ala Trp Asn
340 345 350
Arg Lys Arg Ile Ser Asn Cys Val Ala Asp Tyr Ser Val Leu Tyr Asn
355 360 365
Ser Ala Ser Phe Ser Thr Phe Lys Cys Tyr Gly Val Ser Pro Thr Lys
370 375 380
Leu Asn Asp Leu Cys Phe Thr Asn Val Tyr Ala Asp Ser Phe Val Ile
385 390 395 400
Arg Gly Asp Glu Val Arg Gln Ile Ala Pro Gly Gln Thr Gly Lys Ile
405 410 415
Ala Asp Tyr Asn Tyr Lys Leu Pro Asp Asp Phe Thr Gly Cys Val Ile
420 425 430
Ala Trp Asn Ser Asn Asn Leu Asp Ser Lys Val Gly Gly Asn Tyr Asn
435 440 445
Tyr Arg Tyr Arg Leu Phe Arg Lys Ser Asn Leu Lys Pro Phe Glu Arg
450 455 460
Asp Ile Ser Thr Glu Ile Tyr Gln Ala Gly Ser Lys Pro Cys Asn Gly
465 470 475 480
Val Glu Gly Phe Asn Cys Tyr Phe Pro Leu Gln Ser Tyr Gly Phe Gln
485 490 495
Pro Thr Asn Gly Val Gly Tyr Gln Pro Tyr Arg Val Val Val Leu Ser
500 505 510
Phe Glu Leu Leu His Ala Pro Ala Thr Val Cys Gly Pro Lys Lys Ser
515 520 525
Thr Asn Leu Val Lys Asn Lys Cys Val Asn Phe Asn Phe Asn Gly Leu
530 535 540
Thr Gly Thr Gly Val Leu Thr Glu Ser Asn Lys Lys Phe Leu Pro Phe
545 550 555 560
Gln Gln Phe Gly Arg Asp Ile Ala Asp Thr Thr Asp Ala Val Arg Asp
565 570 575
Pro Gln Thr Leu Glu Ile Leu Asp Ile Thr Pro Cys Ser Phe Gly Gly
580 585 590
Val Ser Val Ile Thr Pro Gly Thr Asn Thr Ser Asn Gln Val Ala Val
595 600 605
Leu Tyr Gln Gly Val Asn Cys Thr Glu Val Pro Val Ala Ile His Ala
610 615 620
Asp Gln Leu Thr Pro Thr Trp Arg Val Tyr Ser Thr Gly Ser Asn Val
625 630 635 640
Phe Gln Thr Arg Ala Gly Cys Leu Ile Gly Ala Glu His Val Asn Asn
645 650 655
Ser Tyr Glu Cys Asp Ile Pro Ile Gly Ala Gly Ile Cys Ala Ser Tyr
660 665 670
Gln Thr Gln Thr Asn Ser Arg Arg Arg Ala Arg Ser Val Ala Ser Gln
675 680 685
Ser Ile Ile Ala Tyr Thr Met Ser Leu Gly Ala Glu Asn Ser Val Ala
690 695 700
Tyr Ser Asn Asn Ser Ile Ala Ile Pro Thr Asn Phe Thr Ile Ser Val
705 710 715 720
Thr Thr Glu Ile Leu Pro Val Ser Met Thr Lys Thr Ser Val Asp Cys
725 730 735
Thr Met Tyr Ile Cys Gly Asp Ser Thr Glu Cys Ser Asn Leu Leu Leu
740 745 750
Gln Tyr Gly Ser Phe Cys Thr Gln Leu Asn Arg Ala Leu Thr Gly Ile
755 760 765
Ala Val Glu Gln Asp Lys Asn Thr Gln Glu Val Phe Ala Gln Val Lys
770 775 780
Gln Ile Tyr Lys Thr Pro Pro Ile Lys Asp Phe Gly Gly Phe Asn Phe
785 790 795 800
Ser Gln Ile Leu Pro Asp Pro Ser Lys Pro Ser Lys Arg Ser Phe Ile
805 810 815
Glu Asp Leu Leu Phe Asn Lys Val Thr Leu Ala Asp Ala Gly Phe Ile
820 825 830
Lys Gln Tyr Gly Asp Cys Leu Gly Asp Ile Ala Ala Arg Asp Leu Ile
835 840 845
Cys Ala Gln Lys Phe Asn Gly Leu Thr Val Leu Pro Pro Leu Leu Thr
850 855 860
Asp Glu Met Ile Ala Gln Tyr Thr Ser Ala Leu Leu Ala Gly Thr Ile
865 870 875 880
Thr Ser Gly Trp Thr Phe Gly Ala Gly Ala Ala Leu Gln Ile Pro Phe
885 890 895
Ala Met Gln Met Ala Tyr Arg Phe Asn Gly Ile Gly Val Thr Gln Asn
900 905 910
Val Leu Tyr Glu Asn Gln Lys Leu Ile Ala Asn Gln Phe Asn Ser Ala
915 920 925
Ile Gly Lys Ile Gln Asp Ser Leu Ser Ser Thr Ala Ser Ala Leu Gly
930 935 940
Lys Leu Gln Asn Val Val Asn Gln Asn Ala Gln Ala Leu Asn Thr Leu
945 950 955 960
Val Lys Gln Leu Ser Ser Asn Phe Gly Ala Ile Ser Ser Val Leu Asn
965 970 975
Asp Ile Leu Ser Arg Leu Asp Lys Val Glu Ala Glu Val Gln Ile Asp
980 985 990
Arg Leu Ile Thr Gly Arg Leu Gln Ser Leu Gln Thr Tyr Val Thr Gln
995 1000 1005
Gln Leu Ile Arg Ala Ala Glu Ile Arg Ala Ser Ala Asn Leu Ala
1010 1015 1020
Ala Thr Lys Met Ser Glu Cys Val Leu Gly Gln Ser Lys Arg Val
1025 1030 1035
Asp Phe Cys Gly Lys Gly Tyr His Leu Met Ser Phe Pro Gln Ser
1040 1045 1050
Ala Pro His Gly Val Val Phe Leu His Val Thr Tyr Val Pro Ala
1055 1060 1065
Gln Glu Lys Asn Phe Thr Thr Ala Pro Ala Ile Cys His Asp Gly
1070 1075 1080
Lys Ala His Phe Pro Arg Glu Gly Val Phe Val Ser Asn Gly Thr
1085 1090 1095
His Trp Phe Val Thr Gln Arg Asn Phe Tyr Glu Pro Gln Ile Ile
1100 1105 1110
Thr Thr Asp Asn Thr Phe Val Ser Gly Asn Cys Asp Val Val Ile
1115 1120 1125
Gly Ile Val Asn Asn Thr Val Tyr Asp Pro Leu Gln Pro Glu Leu
1130 1135 1140
Asp Ser Phe Lys Glu Glu Leu Asp Lys Tyr Phe Lys Asn His Thr
1145 1150 1155
Ser Pro Asp Val Asp Leu Gly Asp Ile Ser Gly Ile Asn Ala Ser
1160 1165 1170
Val Val Asn Ile Gln Lys Glu Ile Asp Arg Leu Asn Glu Val Ala
1175 1180 1185
Lys Asn Leu Asn Glu Ser Leu Ile Asp Leu Gln Glu Leu Gly Lys
1190 1195 1200
Tyr Glu Gln Tyr Ile Lys Trp Pro Trp Tyr Ile Trp Leu Gly Phe
1205 1210 1215
Ile Ala Gly Leu Ile Ala Ile Val Met Val Thr Ile Met Leu Cys
1220 1225 1230
Cys Met Thr Ser Cys Cys Ser Cys Leu Lys Gly Cys Cys Ser Cys
1235 1240 1245
Gly Ser Cys Cys Lys Phe Asp Glu Asp Asp Ser Glu Pro Val Leu
1250 1255 1260
Lys Gly Val Lys Leu His Tyr Thr
1265 1270
<210> 23
<211> 3822
<212> DNA
<213> Artificial Sequence
<220>
<223> S-(deg-RBD) for Wuhan S-2P strain
<400> 23
atgttcgtct tcctggtcct gctgcctctg gtctcctcac agtgcgtcaa tctgacaact 60
cggactcagc tgccacctgc ttatactaat agcttcacca gaggcgtgta ctatcctgac 120
aaggtgttta gaagctccgt gctgcactct acacaggatc tgtttctgcc attctttagc 180
aacgtgacct ggttccacgc catccacgtg agcggcacca atggcacaaa gcggttcgac 240
aatcccgtgc tgccttttaa cgatggcgtg tacttcgcct ctaccgagaa gagcaacatc 300
atcagaggct ggatctttgg caccacactg gactccaaga cacagtctct gctgatcgtg 360
aacaatgcca ccaacgtggt catcaaggtg tgcgagttcc agttttgtaa tgatcccttc 420
ctgggcgtgt actatcacaa gaacaataag agctggatgg agtccgagtt tagagtgtat 480
tctagcgcca acaactgcac atttgagtac gtgagccagc ctttcctgat ggacctggag 540
ggcaagcagg gcaatttcaa gaacctgagg gagttcgtgt ttaagaatat cgacggctac 600
ttcaaaatct actctaagca cacccccatc aacctggtgc gcgacctgcc tcagggcttc 660
agcgccctgg agcccctggt ggatctgcct atcggcatca acatcacccg gtttcagaca 720
ctgctggccc tgcacagaag ctacctgaca cccggcgact cctctagcgg atggaccgcc 780
ggcgctgccg cctactatgt gggctacctc cagccccgga ccttcctgct gaagtacaac 840
gagaatggca ccatcacaga cgcagtggat tgcgccctgg accccctgag cgagacaaag 900
tgtacactga agtcctttac cgtggagaag ggcatctatc agacatccaa tttcagggtg 960
cagccagccg aggcgatcgt gcgctttcct gaaatcacaa acctgtgccc atttggcgag 1020
gtgttcgagg caacccgctt cgccagcgtg tacgcctgga ataggaagcg gatcagcaac 1080
tgcgtggccg actatagcgt gctgtacaac tccgcctctt tcagcacctt taagtgctat 1140
ggcgtgtccc ccacaaagct gaatgacctg tgctttacca acgtctacgc cgattctttc 1200
gtgatcaggg gcgacgaggt gcgccagatc gcccccggcc agacaggcaa gatcgcagac 1260
tacaattata agctgccaga cgatttcacc ggctgcgtga tcgcctggaa cagcaacaat 1320
ctggattcca aagtgggcgg caactacaat tatctgtacc ggctgtttag aaagagcaat 1380
ctgaagccct tcgagaggga catctctaca gaaatctacc aggccggcag caccccttgc 1440
aatggcgtgg agggctttaa ctgttatttc ccactccagt cctacggctt ccagcccaca 1500
aacggcgtgg gctatcagcc ttaccgcgtg gtggtgctga gctttgagct gctgcacgcc 1560
ccagcaacag tgtgcggccc caagaagtcc accaatctgg tgaagaacaa gtgcgtgaac 1620
ttcaacttca acggcctgac cggcacaggc gtgctgaccg agtccaacaa gaagttcctg 1680
ccatttcagc agttcggcag ggacatcgca gataccacag acgccgtgcg cgacccacag 1740
accctggaga tcctggacat cacaccctgc tctttcggcg gcgtgagcgt gatcacaccc 1800
ggcaccaata caagcaacca ggtggccgtg ctgtatcagg acgtgaattg taccgaggtg 1860
cccgtggcta tccacgccga tcagctgacc ccaacatggc gggtgtacag caccggctcc 1920
aacgtcttcc agacaagagc cggatgcctg atcggagcag agcacgtgaa caattcctat 1980
gagtgcgaca tcccaatcgg cgccggcatc tgtgcctctt accagaccca gacaaactct 2040
cccagaagag cccggagcgt ggcctcccag tctatcatcg cctataccat gtccctgggc 2100
gccgagaaca gcgtggccta ctctaacaat agcatcgcca tcccaaccaa cttcacaatc 2160
tctgtgacca cagagatcct gcccgtgtcc atgaccaaga catctgtgga ctgcacaatg 2220
tatatctgtg gcgattctac cgagtgcagc aacctgctgc tccagtacgg cagcttttgt 2280
acccagctga atagagccct gacaggcatc gccgtggagc aggataagaa cacacaggag 2340
gtgttcgccc aggtgaagca aatctacaag acccccccta tcaaggactt tggcggcttc 2400
aatttttccc agatcctgcc tgatccatcc aagccttcta agcggagctt tatcgaggac 2460
ctgctgttca acaaggtgac cctggccgat gccggcttca tcaagcagta tggcgattgc 2520
ctgggcgaca tcgcagccag ggacctgatc tgcgcccaga agtttaatgg cctgaccgtg 2580
ctgccacccc tgctgacaga tgagatgatc gcacagtaca caagcgccct gctggccggc 2640
accatcacat ccggatggac cttcggcgca ggagccgccc tccagatccc ctttgccatg 2700
cagatggcct ataggttcaa cggcatcggc gtgacccaga atgtgctgta cgagaaccag 2760
aagctgatcg ccaatcagtt taactccgcc atcggcaaga tccaggacag cctgtcctct 2820
acagccagcg ccctgggcaa gctccaggat gtggtgaatc agaacgccca ggccctgaat 2880
accctggtga agcagctgag cagcaacttc ggcgccatct ctagcgtgct gaatgacatc 2940
ctgagccggc tggacccgcc ggaggcagag gtgcagatcg accggctgat caccggccgg 3000
ctccagagcc tccagaccta tgtgacacag cagctgatca gggccgccga gatcagggcc 3060
agcgccaatc tggcagcaac caagatgtcc gagtgcgtgc tgggccagtc taagagagtg 3120
gacttttgtg gcaagggcta tcacctgatg tccttccctc agtctgcccc acacggcgtg 3180
gtgtttctgc acgtgaccta cgtgcccgcc caggagaaga acttcaccac agcccctgcc 3240
atctgccacg atggcaaggc ccactttcca agggagggcg tgttcgtgtc caacggcacc 3300
cactggtttg tgacacagcg caatttctac gagccccaga tcatcaccac agacaacacc 3360
ttcgtgagcg gcaactgtga cgtggtcatc ggcatcgtga acaataccgt gtatgatcca 3420
ctccagcccg agctggacag ctttaaggag gagctggata agtatttcaa gaatcacacc 3480
tcccctgacg tggatctggg cgacatcagc ggcatcaatg cctccgtggt gaacatccag 3540
aaggagatcg accgcctgaa cgaggtggct aagaatctga acgagagcct gatcgacctc 3600
caggagctgg gcaagtatga gcagtacatc aagtggccct ggtacatctg gctgggcttc 3660
atcgccggcc tgatcgccat cgtgatggtg accatcatgc tgtgctgtat gacatcctgc 3720
tgttcttgcc tgaagggctg ctgtagctgt ggctcctgct gtaagtttga cgaggatgac 3780
tctgaacctg tgctgaaggg cgtgaagctg cattacacct aa 3822
<210> 24
<211> 1273
<212> PRT
<213> Artificial Sequence
<220>
<223> Protein sequence of S-(deg-RBD) for Wuhan S-2P strain
<400> 24
Met Phe Val Phe Leu Val Leu Leu Pro Leu Val Ser Ser Gln Cys Val
1 5 10 15
Asn Leu Thr Thr Arg Thr Gln Leu Pro Pro Ala Tyr Thr Asn Ser Phe
20 25 30
Thr Arg Gly Val Tyr Tyr Pro Asp Lys Val Phe Arg Ser Ser Val Leu
35 40 45
His Ser Thr Gln Asp Leu Phe Leu Pro Phe Phe Ser Asn Val Thr Trp
50 55 60
Phe His Ala Ile His Val Ser Gly Thr Asn Gly Thr Lys Arg Phe Asp
65 70 75 80
Asn Pro Val Leu Pro Phe Asn Asp Gly Val Tyr Phe Ala Ser Thr Glu
85 90 95
Lys Ser Asn Ile Ile Arg Gly Trp Ile Phe Gly Thr Thr Leu Asp Ser
100 105 110
Lys Thr Gln Ser Leu Leu Ile Val Asn Asn Ala Thr Asn Val Val Ile
115 120 125
Lys Val Cys Glu Phe Gln Phe Cys Asn Asp Pro Phe Leu Gly Val Tyr
130 135 140
Tyr His Lys Asn Asn Lys Ser Trp Met Glu Ser Glu Phe Arg Val Tyr
145 150 155 160
Ser Ser Ala Asn Asn Cys Thr Phe Glu Tyr Val Ser Gln Pro Phe Leu
165 170 175
Met Asp Leu Glu Gly Lys Gln Gly Asn Phe Lys Asn Leu Arg Glu Phe
180 185 190
Val Phe Lys Asn Ile Asp Gly Tyr Phe Lys Ile Tyr Ser Lys His Thr
195 200 205
Pro Ile Asn Leu Val Arg Asp Leu Pro Gln Gly Phe Ser Ala Leu Glu
210 215 220
Pro Leu Val Asp Leu Pro Ile Gly Ile Asn Ile Thr Arg Phe Gln Thr
225 230 235 240
Leu Leu Ala Leu His Arg Ser Tyr Leu Thr Pro Gly Asp Ser Ser Ser
245 250 255
Gly Trp Thr Ala Gly Ala Ala Ala Tyr Tyr Val Gly Tyr Leu Gln Pro
260 265 270
Arg Thr Phe Leu Leu Lys Tyr Asn Glu Asn Gly Thr Ile Thr Asp Ala
275 280 285
Val Asp Cys Ala Leu Asp Pro Leu Ser Glu Thr Lys Cys Thr Leu Lys
290 295 300
Ser Phe Thr Val Glu Lys Gly Ile Tyr Gln Thr Ser Asn Phe Arg Val
305 310 315 320
Gln Pro Ala Glu Ala Ile Val Arg Phe Pro Gln Ile Thr Asn Leu Cys
325 330 335
Pro Phe Gly Glu Val Phe Gln Ala Thr Arg Phe Ala Ser Val Tyr Ala
340 345 350
Trp Asn Arg Lys Arg Ile Ser Asn Cys Val Ala Asp Tyr Ser Val Leu
355 360 365
Tyr Asn Ser Ala Ser Phe Ser Thr Phe Lys Cys Tyr Gly Val Ser Pro
370 375 380
Thr Lys Leu Asn Asp Leu Cys Phe Thr Asn Val Tyr Ala Asp Ser Phe
385 390 395 400
Val Ile Arg Gly Asp Glu Val Arg Gln Ile Ala Pro Gly Gln Thr Gly
405 410 415
Lys Ile Ala Asp Tyr Asn Tyr Lys Leu Pro Asp Asp Phe Thr Gly Cys
420 425 430
Val Ile Ala Trp Asn Ser Asn Asn Leu Asp Ser Lys Val Gly Gly Asn
435 440 445
Tyr Asn Tyr Leu Tyr Arg Leu Phe Arg Lys Ser Asn Leu Lys Pro Phe
450 455 460
Glu Arg Asp Ile Ser Thr Glu Ile Tyr Gln Ala Gly Ser Thr Pro Cys
465 470 475 480
Asn Gly Val Glu Gly Phe Asn Cys Tyr Phe Pro Leu Gln Ser Tyr Gly
485 490 495
Phe Gln Pro Thr Asn Gly Val Gly Tyr Gln Pro Tyr Arg Val Val Val
500 505 510
Leu Ser Phe Glu Leu Leu His Ala Pro Ala Thr Val Cys Gly Pro Lys
515 520 525
Lys Ser Thr Asn Leu Val Lys Asn Lys Cys Val Asn Phe Asn Phe Asn
530 535 540
Gly Leu Thr Gly Thr Gly Val Leu Thr Glu Ser Asn Lys Lys Phe Leu
545 550 555 560
Pro Phe Gln Gln Phe Gly Arg Asp Ile Ala Asp Thr Thr Asp Ala Val
565 570 575
Arg Asp Pro Gln Thr Leu Glu Ile Leu Asp Ile Thr Pro Cys Ser Phe
580 585 590
Gly Gly Val Ser Val Ile Thr Pro Gly Thr Asn Thr Ser Asn Gln Val
595 600 605
Ala Val Leu Tyr Gln Asp Val Asn Cys Thr Glu Val Pro Val Ala Ile
610 615 620
His Ala Asp Gln Leu Thr Pro Thr Trp Arg Val Tyr Ser Thr Gly Ser
625 630 635 640
Asn Val Phe Gln Thr Arg Ala Gly Cys Leu Ile Gly Ala Glu His Val
645 650 655
Asn Asn Ser Tyr Glu Cys Asp Ile Pro Ile Gly Ala Gly Ile Cys Ala
660 665 670
Ser Tyr Gln Thr Gln Thr Asn Ser Pro Arg Arg Ala Arg Ser Val Ala
675 680 685
Ser Gln Ser Ile Ile Ala Tyr Thr Met Ser Leu Gly Ala Glu Asn Ser
690 695 700
Val Ala Tyr Ser Asn Asn Ser Ile Ala Ile Pro Thr Asn Phe Thr Ile
705 710 715 720
Ser Val Thr Thr Glu Ile Leu Pro Val Ser Met Thr Lys Thr Ser Val
725 730 735
Asp Cys Thr Met Tyr Ile Cys Gly Asp Ser Thr Glu Cys Ser Asn Leu
740 745 750
Leu Leu Gln Tyr Gly Ser Phe Cys Thr Gln Leu Asn Arg Ala Leu Thr
755 760 765
Gly Ile Ala Val Glu Gln Asp Lys Asn Thr Gln Glu Val Phe Ala Gln
770 775 780
Val Lys Gln Ile Tyr Lys Thr Pro Pro Ile Lys Asp Phe Gly Gly Phe
785 790 795 800
Asn Phe Ser Gln Ile Leu Pro Asp Pro Ser Lys Pro Ser Lys Arg Ser
805 810 815
Phe Ile Glu Asp Leu Leu Phe Asn Lys Val Thr Leu Ala Asp Ala Gly
820 825 830
Phe Ile Lys Gln Tyr Gly Asp Cys Leu Gly Asp Ile Ala Ala Arg Asp
835 840 845
Leu Ile Cys Ala Gln Lys Phe Asn Gly Leu Thr Val Leu Pro Pro Leu
850 855 860
Leu Thr Asp Glu Met Ile Ala Gln Tyr Thr Ser Ala Leu Leu Ala Gly
865 870 875 880
Thr Ile Thr Ser Gly Trp Thr Phe Gly Ala Gly Ala Ala Leu Gln Ile
885 890 895
Pro Phe Ala Met Gln Met Ala Tyr Arg Phe Asn Gly Ile Gly Val Thr
900 905 910
Gln Asn Val Leu Tyr Glu Asn Gln Lys Leu Ile Ala Asn Gln Phe Asn
915 920 925
Ser Ala Ile Gly Lys Ile Gln Asp Ser Leu Ser Ser Thr Ala Ser Ala
930 935 940
Leu Gly Lys Leu Gln Asp Val Val Asn Gln Asn Ala Gln Ala Leu Asn
945 950 955 960
Thr Leu Val Lys Gln Leu Ser Ser Asn Phe Gly Ala Ile Ser Ser Val
965 970 975
Leu Asn Asp Ile Leu Ser Arg Leu Asp Pro Pro Glu Ala Glu Val Gln
980 985 990
Ile Asp Arg Leu Ile Thr Gly Arg Leu Gln Ser Leu Gln Thr Tyr Val
995 1000 1005
Thr Gln Gln Leu Ile Arg Ala Ala Glu Ile Arg Ala Ser Ala Asn
1010 1015 1020
Leu Ala Ala Thr Lys Met Ser Glu Cys Val Leu Gly Gln Ser Lys
1025 1030 1035
Arg Val Asp Phe Cys Gly Lys Gly Tyr His Leu Met Ser Phe Pro
1040 1045 1050
Gln Ser Ala Pro His Gly Val Val Phe Leu His Val Thr Tyr Val
1055 1060 1065
Pro Ala Gln Glu Lys Asn Phe Thr Thr Ala Pro Ala Ile Cys His
1070 1075 1080
Asp Gly Lys Ala His Phe Pro Arg Glu Gly Val Phe Val Ser Asn
1085 1090 1095
Gly Thr His Trp Phe Val Thr Gln Arg Asn Phe Tyr Glu Pro Gln
1100 1105 1110
Ile Ile Thr Thr Asp Asn Thr Phe Val Ser Gly Asn Cys Asp Val
1115 1120 1125
Val Ile Gly Ile Val Asn Asn Thr Val Tyr Asp Pro Leu Gln Pro
1130 1135 1140
Glu Leu Asp Ser Phe Lys Glu Glu Leu Asp Lys Tyr Phe Lys Asn
1145 1150 1155
His Thr Ser Pro Asp Val Asp Leu Gly Asp Ile Ser Gly Ile Asn
1160 1165 1170
Ala Ser Val Val Asn Ile Gln Lys Glu Ile Asp Arg Leu Asn Glu
1175 1180 1185
Val Ala Lys Asn Leu Asn Glu Ser Leu Ile Asp Leu Gln Glu Leu
1190 1195 1200
Gly Lys Tyr Glu Gln Tyr Ile Lys Trp Pro Trp Tyr Ile Trp Leu
1205 1210 1215
Gly Phe Ile Ala Gly Leu Ile Ala Ile Val Met Val Thr Ile Met
1220 1225 1230
Leu Cys Cys Met Thr Ser Cys Cys Ser Cys Leu Lys Gly Cys Cys
1235 1240 1245
Ser Cys Gly Ser Cys Cys Lys Phe Asp Glu Asp Asp Ser Glu Pro
1250 1255 1260
Val Leu Lys Gly Val Lys Leu His Tyr Thr
1265 1270
<210> 25
<211> 3816
<212> DNA
<213> Artificial Sequence
<220>
<223> S-(deg-RBD) for Delta S-2P strain
<400> 25
atgttcgtct tcctggtcct gctgcctctg gtctcctcac agtgcgtcaa tctgcgaact 60
cggactcagc tgccacctgc ttatactaat agcttcacca gaggcgtgta ctatcctgac 120
aaggtgttta gaagctccgt gctgcactct acacaggatc tgtttctgcc attctttagc 180
aacgtgacct ggttccacgc catccacgtg agcggcacca atggcacaaa gcggttcgac 240
aatcccgtgc tgccttttaa cgatggcgtg tacttcgcct ctatcgagaa gagcaacatc 300
atcagaggct ggatctttgg caccacactg gactccaaga cacagtctct gctgatcgtg 360
aacaatgcca ccaacgtggt catcaaggtg tgcgagttcc agttttgtaa tgatcccttc 420
ctggacgtgt actatcacaa gaacaataag agctggatgg agtccggagt gtattctagc 480
gccaacaact gcacatttga gtacgtgagc cagcctttcc tgatggacct ggagggcaag 540
cagggcaatt tcaagaacct gagggagttc gtgtttaaga atatcgacgg ctacttcaaa 600
atctactcta agcacacccc catcaacctg gtgcgcgacc tgcctcaggg cttcagcgcc 660
ctggagcccc tggtggatct gcctatcggc atcaacatca cccggtttca gacactgctg 720
gccctgcaca gaagctacct gacacccggc gactcctcta gcggatggac cgccggcgct 780
gccgcctact atgtgggcta cctccagccc cggaccttcc tgctgaagta caacgagaat 840
ggcaccatca cagacgcagt ggattgcgcc ctggaccccc tgagcgagac aaagtgtaca 900
ctgaagtcct ttaccgtgga gaagggcatc tatcagacat ccaatttcag ggtgcagcca 960
gccgaggcga tcgtgcgctt tcctgaaatc acaaacctgt gcccatttgg cgaggtgttc 1020
gaggcaaccc gcttcgccag cgtgtacgcc tggaatagga agcggatcag caactgcgtg 1080
gccgactata gcgtgctgta caactccgcc tctttcagca cctttaagtg ctatggcgtg 1140
tcccccacaa agctgaatga cctgtgcttt accaacgtct acgccgattc tttcgtgatc 1200
aggggcgacg aggtgcgcca gatcgccccc ggccagacag gcaagatcgc agactacaat 1260
tataagctgc cagacgattt caccggctgc gtgatcgcct ggaacagcaa caatctggat 1320
tccaaagtgg gcggcaacta caattatcgg taccggctgt ttagaaagag caatctgaag 1380
cccttcgaga gggacatctc tacagaaatc taccaggccg gcagcaagcc ttgcaatggc 1440
gtggagggct ttaactgtta tttcccactc cagtcctacg gcttccagcc cacaaacggc 1500
gtgggctatc agccttaccg cgtggtggtg ctgagctttg agctgctgca cgccccagca 1560
acagtgtgcg gccccaagaa gtccaccaat ctggtgaaga acaagtgcgt gaacttcaac 1620
ttcaacggcc tgaccggcac aggcgtgctg accgagtcca acaagaagtt cctgccattt 1680
cagcagttcg gcagggacat cgcagatacc acagacgccg tgcgcgaccc acagaccctg 1740
gagatcctgg acatcacacc ctgctctttc ggcggcgtga gcgtgatcac acccggcacc 1800
aatacaagca accaggtggc cgtgctgtat cagggcgtga attgtaccga ggtgcccgtg 1860
gctatccacg ccgatcagct gaccccaaca tggcgggtgt acagcaccgg ctccaacgtc 1920
ttccagacaa gagccggatg cctgatcgga gcagagcacg tgaacaattc ctatgagtgc 1980
gacatcccaa tcggcgccgg catctgtgcc tcttaccaga cccagacaaa ctctcgcaga 2040
agagcccgga gcgtggcctc ccagtctatc atcgcctata ccatgtccct gggcgccgag 2100
aacagcgtgg cctactctaa caatagcatc gccatcccaa ccaacttcac aatctctgtg 2160
accacagaga tcctgcccgt gtccatgacc aagacatctg tggactgcac aatgtatatc 2220
tgtggcgatt ctaccgagtg cagcaacctg ctgctccagt acggcagctt ttgtacccag 2280
ctgaatagag ccctgacagg catcgccgtg gagcaggata agaacacaca ggaggtgttc 2340
gcccaggtga agcaaatcta caagaccccc cctatcaagg actttggcgg cttcaatttt 2400
tcccagatcc tgcctgatcc atccaagcct tctaagcgga gctttatcga ggacctgctg 2460
ttcaacaagg tgaccctggc cgatgccggc ttcatcaagc agtatggcga ttgcctgggc 2520
gacatcgcag ccagggacct gatctgcgcc cagaagttta atggcctgac cgtgctgcca 2580
cccctgctga cagatgagat gatcgcacag tacacaagcg ccctgctggc cggcaccatc 2640
acatccggat ggaccttcgg cgcaggagcc gccctccaga tcccctttgc catgcagatg 2700
gcctataggt tcaacggcat cggcgtgacc cagaatgtgc tgtacgagaa ccagaagctg 2760
atcgccaatc agtttaactc cgccatcggc aagatccagg acagcctgtc ctctacagcc 2820
agcgccctgg gcaagctcca gaatgtggtg aatcagaacg cccaggccct gaataccctg 2880
gtgaagcagc tgagcagcaa cttcggcgcc atctctagcg tgctgaatga catcctgagc 2940
cggctggacc cgccggaggc agaggtgcag atcgaccggc tgatcaccgg ccggctccag 3000
agcctccaga cctatgtgac acagcagctg atcagggccg ccgagatcag ggccagcgcc 3060
aatctggcag caaccaagat gtccgagtgc gtgctgggcc agtctaagag agtggacttt 3120
tgtggcaagg gctatcacct gatgtccttc cctcagtctg ccccacacgg cgtggtgttt 3180
ctgcacgtga cctacgtgcc cgcccaggag aagaacttca ccacagcccc tgccatctgc 3240
cacgatggca aggcccactt tccaagggag ggcgtgttcg tgtccaacgg cacccactgg 3300
tttgtgacac agcgcaattt ctacgagccc cagatcatca ccacagacaa caccttcgtg 3360
agcggcaact gtgacgtggt catcggcatc gtgaacaata ccgtgtatga tccactccag 3420
cccgagctgg acagctttaa ggaggagctg gataagtatt tcaagaatca cacctcccct 3480
gacgtggatc tgggcgacat cagcggcatc aatgcctccg tggtgaacat ccagaaggag 3540
atcgaccgcc tgaacgaggt ggctaagaat ctgaacgaga gcctgatcga cctccaggag 3600
ctgggcaagt atgagcagta catcaagtgg ccctggtaca tctggctggg cttcatcgcc 3660
ggcctgatcg ccatcgtgat ggtgaccatc atgctgtgct gtatgacatc ctgctgttct 3720
tgcctgaagg gctgctgtag ctgtggctcc tgctgtaagt ttgacgagga tgactctgaa 3780
cctgtgctga agggcgtgaa gctgcattac acctaa 3816
<210> 26
<211> 1271
<212> PRT
<213> Artificial Sequence
<220>
<223> S-(deg-RBD) for Delta S-2P strain
<400> 26
Met Phe Val Phe Leu Val Leu Leu Pro Leu Val Ser Ser Gln Cys Val
1 5 10 15
Asn Leu Arg Thr Arg Thr Gln Leu Pro Pro Ala Tyr Thr Asn Ser Phe
20 25 30
Thr Arg Gly Val Tyr Tyr Pro Asp Lys Val Phe Arg Ser Ser Val Leu
35 40 45
His Ser Thr Gln Asp Leu Phe Leu Pro Phe Phe Ser Asn Val Thr Trp
50 55 60
Phe His Ala Ile His Val Ser Gly Thr Asn Gly Thr Lys Arg Phe Asp
65 70 75 80
Asn Pro Val Leu Pro Phe Asn Asp Gly Val Tyr Phe Ala Ser Ile Glu
85 90 95
Lys Ser Asn Ile Ile Arg Gly Trp Ile Phe Gly Thr Thr Leu Asp Ser
100 105 110
Lys Thr Gln Ser Leu Leu Ile Val Asn Asn Ala Thr Asn Val Val Ile
115 120 125
Lys Val Cys Glu Phe Gln Phe Cys Asn Asp Pro Phe Leu Asp Val Tyr
130 135 140
Tyr His Lys Asn Asn Lys Ser Trp Met Glu Ser Gly Val Tyr Ser Ser
145 150 155 160
Ala Asn Asn Cys Thr Phe Glu Tyr Val Ser Gln Pro Phe Leu Met Asp
165 170 175
Leu Glu Gly Lys Gln Gly Asn Phe Lys Asn Leu Arg Glu Phe Val Phe
180 185 190
Lys Asn Ile Asp Gly Tyr Phe Lys Ile Tyr Ser Lys His Thr Pro Ile
195 200 205
Asn Leu Val Arg Asp Leu Pro Gln Gly Phe Ser Ala Leu Glu Pro Leu
210 215 220
Val Asp Leu Pro Ile Gly Ile Asn Ile Thr Arg Phe Gln Thr Leu Leu
225 230 235 240
Ala Leu His Arg Ser Tyr Leu Thr Pro Gly Asp Ser Ser Ser Gly Trp
245 250 255
Thr Ala Gly Ala Ala Ala Tyr Tyr Val Gly Tyr Leu Gln Pro Arg Thr
260 265 270
Phe Leu Leu Lys Tyr Asn Glu Asn Gly Thr Ile Thr Asp Ala Val Asp
275 280 285
Cys Ala Leu Asp Pro Leu Ser Glu Thr Lys Cys Thr Leu Lys Ser Phe
290 295 300
Thr Val Glu Lys Gly Ile Tyr Gln Thr Ser Asn Phe Arg Val Gln Pro
305 310 315 320
Ala Glu Ala Ile Val Arg Phe Pro Gln Ile Thr Asn Leu Cys Pro Phe
325 330 335
Gly Glu Val Phe Gln Ala Thr Arg Phe Ala Ser Val Tyr Ala Trp Asn
340 345 350
Arg Lys Arg Ile Ser Asn Cys Val Ala Asp Tyr Ser Val Leu Tyr Asn
355 360 365
Ser Ala Ser Phe Ser Thr Phe Lys Cys Tyr Gly Val Ser Pro Thr Lys
370 375 380
Leu Asn Asp Leu Cys Phe Thr Asn Val Tyr Ala Asp Ser Phe Val Ile
385 390 395 400
Arg Gly Asp Glu Val Arg Gln Ile Ala Pro Gly Gln Thr Gly Lys Ile
405 410 415
Ala Asp Tyr Asn Tyr Lys Leu Pro Asp Asp Phe Thr Gly Cys Val Ile
420 425 430
Ala Trp Asn Ser Asn Asn Leu Asp Ser Lys Val Gly Gly Asn Tyr Asn
435 440 445
Tyr Arg Tyr Arg Leu Phe Arg Lys Ser Asn Leu Lys Pro Phe Glu Arg
450 455 460
Asp Ile Ser Thr Glu Ile Tyr Gln Ala Gly Ser Lys Pro Cys Asn Gly
465 470 475 480
Val Glu Gly Phe Asn Cys Tyr Phe Pro Leu Gln Ser Tyr Gly Phe Gln
485 490 495
Pro Thr Asn Gly Val Gly Tyr Gln Pro Tyr Arg Val Val Val Leu Ser
500 505 510
Phe Glu Leu Leu His Ala Pro Ala Thr Val Cys Gly Pro Lys Lys Ser
515 520 525
Thr Asn Leu Val Lys Asn Lys Cys Val Asn Phe Asn Phe Asn Gly Leu
530 535 540
Thr Gly Thr Gly Val Leu Thr Glu Ser Asn Lys Lys Phe Leu Pro Phe
545 550 555 560
Gln Gln Phe Gly Arg Asp Ile Ala Asp Thr Thr Asp Ala Val Arg Asp
565 570 575
Pro Gln Thr Leu Glu Ile Leu Asp Ile Thr Pro Cys Ser Phe Gly Gly
580 585 590
Val Ser Val Ile Thr Pro Gly Thr Asn Thr Ser Asn Gln Val Ala Val
595 600 605
Leu Tyr Gln Gly Val Asn Cys Thr Glu Val Pro Val Ala Ile His Ala
610 615 620
Asp Gln Leu Thr Pro Thr Trp Arg Val Tyr Ser Thr Gly Ser Asn Val
625 630 635 640
Phe Gln Thr Arg Ala Gly Cys Leu Ile Gly Ala Glu His Val Asn Asn
645 650 655
Ser Tyr Glu Cys Asp Ile Pro Ile Gly Ala Gly Ile Cys Ala Ser Tyr
660 665 670
Gln Thr Gln Thr Asn Ser Arg Arg Arg Ala Arg Ser Val Ala Ser Gln
675 680 685
Ser Ile Ile Ala Tyr Thr Met Ser Leu Gly Ala Glu Asn Ser Val Ala
690 695 700
Tyr Ser Asn Asn Ser Ile Ala Ile Pro Thr Asn Phe Thr Ile Ser Val
705 710 715 720
Thr Thr Glu Ile Leu Pro Val Ser Met Thr Lys Thr Ser Val Asp Cys
725 730 735
Thr Met Tyr Ile Cys Gly Asp Ser Thr Glu Cys Ser Asn Leu Leu Leu
740 745 750
Gln Tyr Gly Ser Phe Cys Thr Gln Leu Asn Arg Ala Leu Thr Gly Ile
755 760 765
Ala Val Glu Gln Asp Lys Asn Thr Gln Glu Val Phe Ala Gln Val Lys
770 775 780
Gln Ile Tyr Lys Thr Pro Pro Ile Lys Asp Phe Gly Gly Phe Asn Phe
785 790 795 800
Ser Gln Ile Leu Pro Asp Pro Ser Lys Pro Ser Lys Arg Ser Phe Ile
805 810 815
Glu Asp Leu Leu Phe Asn Lys Val Thr Leu Ala Asp Ala Gly Phe Ile
820 825 830
Lys Gln Tyr Gly Asp Cys Leu Gly Asp Ile Ala Ala Arg Asp Leu Ile
835 840 845
Cys Ala Gln Lys Phe Asn Gly Leu Thr Val Leu Pro Pro Leu Leu Thr
850 855 860
Asp Glu Met Ile Ala Gln Tyr Thr Ser Ala Leu Leu Ala Gly Thr Ile
865 870 875 880
Thr Ser Gly Trp Thr Phe Gly Ala Gly Ala Ala Leu Gln Ile Pro Phe
885 890 895
Ala Met Gln Met Ala Tyr Arg Phe Asn Gly Ile Gly Val Thr Gln Asn
900 905 910
Val Leu Tyr Glu Asn Gln Lys Leu Ile Ala Asn Gln Phe Asn Ser Ala
915 920 925
Ile Gly Lys Ile Gln Asp Ser Leu Ser Ser Thr Ala Ser Ala Leu Gly
930 935 940
Lys Leu Gln Asn Val Val Asn Gln Asn Ala Gln Ala Leu Asn Thr Leu
945 950 955 960
Val Lys Gln Leu Ser Ser Asn Phe Gly Ala Ile Ser Ser Val Leu Asn
965 970 975
Asp Ile Leu Ser Arg Leu Asp Pro Pro Glu Ala Glu Val Gln Ile Asp
980 985 990
Arg Leu Ile Thr Gly Arg Leu Gln Ser Leu Gln Thr Tyr Val Thr Gln
995 1000 1005
Gln Leu Ile Arg Ala Ala Glu Ile Arg Ala Ser Ala Asn Leu Ala
1010 1015 1020
Ala Thr Lys Met Ser Glu Cys Val Leu Gly Gln Ser Lys Arg Val
1025 1030 1035
Asp Phe Cys Gly Lys Gly Tyr His Leu Met Ser Phe Pro Gln Ser
1040 1045 1050
Ala Pro His Gly Val Val Phe Leu His Val Thr Tyr Val Pro Ala
1055 1060 1065
Gln Glu Lys Asn Phe Thr Thr Ala Pro Ala Ile Cys His Asp Gly
1070 1075 1080
Lys Ala His Phe Pro Arg Glu Gly Val Phe Val Ser Asn Gly Thr
1085 1090 1095
His Trp Phe Val Thr Gln Arg Asn Phe Tyr Glu Pro Gln Ile Ile
1100 1105 1110
Thr Thr Asp Asn Thr Phe Val Ser Gly Asn Cys Asp Val Val Ile
1115 1120 1125
Gly Ile Val Asn Asn Thr Val Tyr Asp Pro Leu Gln Pro Glu Leu
1130 1135 1140
Asp Ser Phe Lys Glu Glu Leu Asp Lys Tyr Phe Lys Asn His Thr
1145 1150 1155
Ser Pro Asp Val Asp Leu Gly Asp Ile Ser Gly Ile Asn Ala Ser
1160 1165 1170
Val Val Asn Ile Gln Lys Glu Ile Asp Arg Leu Asn Glu Val Ala
1175 1180 1185
Lys Asn Leu Asn Glu Ser Leu Ile Asp Leu Gln Glu Leu Gly Lys
1190 1195 1200
Tyr Glu Gln Tyr Ile Lys Trp Pro Trp Tyr Ile Trp Leu Gly Phe
1205 1210 1215
Ile Ala Gly Leu Ile Ala Ile Val Met Val Thr Ile Met Leu Cys
1220 1225 1230
Cys Met Thr Ser Cys Cys Ser Cys Leu Lys Gly Cys Cys Ser Cys
1235 1240 1245
Gly Ser Cys Cys Lys Phe Asp Glu Asp Asp Ser Glu Pro Val Leu
1250 1255 1260
Lys Gly Val Lys Leu His Tyr Thr
1265 1270
<210> 27
<211> 3816
<212> DNA
<213> Artificial Sequence
<220>
<223> S-(deg-S2) for Delta strain
<400> 27
atgttcgtct tcctggtcct gctgcctctg gtctcctcac agtgcgtcaa tctgcgaact 60
cggactcagc tgccacctgc ttatactaat agcttcacca gaggcgtgta ctatcctgac 120
aaggtgttta gaagctccgt gctgcactct acacaggatc tgtttctgcc attctttagc 180
aacgtgacct ggttccacgc catccacgtg agcggcacca atggcacaaa gcggttcgac 240
aatcccgtgc tgccttttaa cgatggcgtg tacttcgcct ctatcgagaa gagcaacatc 300
atcagaggct ggatctttgg caccacactg gactccaaga cacagtctct gctgatcgtg 360
aacaatgcca ccaacgtggt catcaaggtg tgcgagttcc agttttgtaa tgatcccttc 420
ctggacgtgt actatcacaa gaacaataag agctggatgg agtccggagt gtattctagc 480
gccaacaact gcacatttga gtacgtgagc cagcctttcc tgatggacct ggagggcaag 540
cagggcaatt tcaagaacct gagggagttc gtgtttaaga atatcgacgg ctacttcaaa 600
atctactcta agcacacccc catcaacctg gtgcgcgacc tgcctcaggg cttcagcgcc 660
ctggagcccc tggtggatct gcctatcggc atcaacatca cccggtttca gacactgctg 720
gccctgcaca gaagctacct gacacccggc gactcctcta gcggatggac cgccggcgct 780
gccgcctact atgtgggcta cctccagccc cggaccttcc tgctgaagta caacgagaat 840
ggcaccatca cagacgcagt ggattgcgcc ctggaccccc tgagcgagac aaagtgtaca 900
ctgaagtcct ttaccgtgga gaagggcatc tatcagacat ccaatttcag ggtgcagcca 960
accgagtcta tcgtgcgctt tcctaatatc acaaacctgt gcccatttgg cgaggtgttc 1020
aacgcaaccc gcttcgccag cgtgtacgcc tggaatagga agcggatcag caactgcgtg 1080
gccgactata gcgtgctgta caactccgcc tctttcagca cctttaagtg ctatggcgtg 1140
tcccccacaa agctgaatga cctgtgcttt accaacgtct acgccgattc tttcgtgatc 1200
aggggcgacg aggtgcgcca gatcgccccc ggccagacag gcaagatcgc agactacaat 1260
tataagctgc cagacgattt caccggctgc gtgatcgcct ggaacagcaa caatctggat 1320
tccaaagtgg gcggcaacta caattatcgg taccggctgt ttagaaagag caatctgaag 1380
cccttcgaga gggacatctc tacagaaatc taccaggccg gcagcaagcc ttgcaatggc 1440
gtggagggct ttaactgtta tttcccactc cagtcctacg gcttccagcc cacaaacggc 1500
gtgggctatc agccttaccg cgtggtggtg ctgagctttg agctgctgca cgccccagca 1560
acagtgtgcg gccccaagaa gtccaccaat ctggtgaaga acaagtgcgt gaacttcaac 1620
ttcaacggcc tgaccggcac aggcgtgctg accgagtcca acaagaagtt cctgccattt 1680
cagcagttcg gcagggacat cgcagatacc acagacgccg tgcgcgaccc acagaccctg 1740
gagatcctgg acatcacacc ctgctctttc ggcggcgtga gcgtgatcac acccggcacc 1800
aatacaagca accaggtggc cgtgctgtat cagggcgtga attgtaccga ggtgcccgtg 1860
gctatccacg ccgatcagct gaccccaaca tggcgggtgt acagcaccgg ctccaacgtc 1920
ttccagacaa gagccggatg cctgatcgga gcagagcacg tgaacaattc ctatgagtgc 1980
gacatcccaa tcggcgccgg catctgtgcc tcttaccaga cccagacaaa ctctcgcaga 2040
agagcccgga gcgtggcctc ccagtctatc atcgcctata ccatgtccct gggcgccgag 2100
aacagcgtgg cctactctca gaatagcatc gccatcccaa cccagttcac aatctctgtg 2160
accacagaga tcctgcccgt gtccatgacc aagacatctg tggactgcac aatgtatatc 2220
tgtggcgatt ctaccgagtg cagcaacctg ctgctccagt acggcagctt ttgtacccag 2280
ctgaatagag ccctgacagg catcgccgtg gagcaggata agaacacaca ggaggtgttc 2340
gcccaggtga agcaaatcta caagaccccc cctatcaagg actttggcgg cttccaattt 2400
tcccagatcc tgcctgatcc atccaagcct tctaagcgga gctttatcga ggacctgctg 2460
ttcaacaagg tgaccctggc cgatgccggc ttcatcaagc agtatggcga ttgcctgggc 2520
gacatcgcag ccagggacct gatctgcgcc cagaagttta atggcctgac cgtgctgcca 2580
cccctgctga cagatgagat gatcgcacag tacacaagcg ccctgctggc cggcaccatc 2640
acatccggat ggaccttcgg cgcaggagcc gccctccaga tcccctttgc catgcagatg 2700
gcctataggt tcaacggcat cggcgtgacc cagaatgtgc tgtacgagaa ccagaagctg 2760
atcgccaatc agtttaactc cgccatcggc aagatccagg acagcctgtc ctctacagcc 2820
agcgccctgg gcaagctcca gaatgtggtg aatcagaacg cccaggccct gaataccctg 2880
gtgaagcagc tgagcagcaa cttcggcgcc atctctagcg tgctgaatga catcctgagc 2940
cggctggaca aggtggaggc agaggtgcag atcgaccggc tgatcaccgg ccggctccag 3000
agcctccaga cctatgtgac acagcagctg atcagggccg ccgagatcag ggccagcgcc 3060
aatctggcag caaccaagat gtccgagtgc gtgctgggcc agtctaagag agtggacttt 3120
tgtggcaagg gctatcacct gatgtccttc cctcagtctg ccccacacgg cgtggtgttt 3180
ctgcacgtga cctacgtgcc cgcccaggag aagcagttca ccacagcccc tgccatctgc 3240
cacgatggca aggcccactt tccaagggag ggcgtgttcg tgtcccaggg cacccactgg 3300
tttgtgacac agcgcaattt ctacgagccc cagatcatca ccacagacaa caccttcgtg 3360
agcggcaact gtgacgtggt catcggcatc gtgcagaata ccgtgtatga tccactccag 3420
cccgagctgg acagctttaa ggaggagctg gataagtatt tcaagcaaca cacctcccct 3480
gacgtggatc tgggcgacat cagcggcatc caagcctccg tggtgaacat ccagaaggag 3540
atcgaccgcc tgaacgaggt ggctaagaat ctgcaggaga gcctgatcga cctccaggag 3600
ctgggcaagt atgagcagta catcaagtgg ccctggtaca tctggctggg cttcatcgcc 3660
ggcctgatcg ccatcgtgat ggtgaccatc atgctgtgct gtatgacatc ctgctgttct 3720
tgcctgaagg gctgctgtag ctgtggctcc tgctgtaagt ttgacgagga tgactctgaa 3780
cctgtgctga agggcgtgaa gctgcattac acctaa 3816
<210> 28
<211> 1271
<212> PRT
<213> Artificial Sequence
<220>
<223> S-(deg-S2) for Delta strain
<400> 28
Met Phe Val Phe Leu Val Leu Leu Pro Leu Val Ser Ser Gln Cys Val
1 5 10 15
Asn Leu Arg Thr Arg Thr Gln Leu Pro Pro Ala Tyr Thr Asn Ser Phe
20 25 30
Thr Arg Gly Val Tyr Tyr Pro Asp Lys Val Phe Arg Ser Ser Val Leu
35 40 45
His Ser Thr Gln Asp Leu Phe Leu Pro Phe Phe Ser Asn Val Thr Trp
50 55 60
Phe His Ala Ile His Val Ser Gly Thr Asn Gly Thr Lys Arg Phe Asp
65 70 75 80
Asn Pro Val Leu Pro Phe Asn Asp Gly Val Tyr Phe Ala Ser Ile Glu
85 90 95
Lys Ser Asn Ile Ile Arg Gly Trp Ile Phe Gly Thr Thr Leu Asp Ser
100 105 110
Lys Thr Gln Ser Leu Leu Ile Val Asn Asn Ala Thr Asn Val Val Ile
115 120 125
Lys Val Cys Glu Phe Gln Phe Cys Asn Asp Pro Phe Leu Asp Val Tyr
130 135 140
Tyr His Lys Asn Asn Lys Ser Trp Met Glu Ser Gly Val Tyr Ser Ser
145 150 155 160
Ala Asn Asn Cys Thr Phe Glu Tyr Val Ser Gln Pro Phe Leu Met Asp
165 170 175
Leu Glu Gly Lys Gln Gly Asn Phe Lys Asn Leu Arg Glu Phe Val Phe
180 185 190
Lys Asn Ile Asp Gly Tyr Phe Lys Ile Tyr Ser Lys His Thr Pro Ile
195 200 205
Asn Leu Val Arg Asp Leu Pro Gln Gly Phe Ser Ala Leu Glu Pro Leu
210 215 220
Val Asp Leu Pro Ile Gly Ile Asn Ile Thr Arg Phe Gln Thr Leu Leu
225 230 235 240
Ala Leu His Arg Ser Tyr Leu Thr Pro Gly Asp Ser Ser Ser Gly Trp
245 250 255
Thr Ala Gly Ala Ala Ala Tyr Tyr Val Gly Tyr Leu Gln Pro Arg Thr
260 265 270
Phe Leu Leu Lys Tyr Asn Glu Asn Gly Thr Ile Thr Asp Ala Val Asp
275 280 285
Cys Ala Leu Asp Pro Leu Ser Glu Thr Lys Cys Thr Leu Lys Ser Phe
290 295 300
Thr Val Glu Lys Gly Ile Tyr Gln Thr Ser Asn Phe Arg Val Gln Pro
305 310 315 320
Thr Glu Ser Ile Val Arg Phe Pro Asn Ile Thr Asn Leu Cys Pro Phe
325 330 335
Gly Glu Val Phe Asn Ala Thr Arg Phe Ala Ser Val Tyr Ala Trp Asn
340 345 350
Arg Lys Arg Ile Ser Asn Cys Val Ala Asp Tyr Ser Val Leu Tyr Asn
355 360 365
Ser Ala Ser Phe Ser Thr Phe Lys Cys Tyr Gly Val Ser Pro Thr Lys
370 375 380
Leu Asn Asp Leu Cys Phe Thr Asn Val Tyr Ala Asp Ser Phe Val Ile
385 390 395 400
Arg Gly Asp Glu Val Arg Gln Ile Ala Pro Gly Gln Thr Gly Lys Ile
405 410 415
Ala Asp Tyr Asn Tyr Lys Leu Pro Asp Asp Phe Thr Gly Cys Val Ile
420 425 430
Ala Trp Asn Ser Asn Asn Leu Asp Ser Lys Val Gly Gly Asn Tyr Asn
435 440 445
Tyr Arg Tyr Arg Leu Phe Arg Lys Ser Asn Leu Lys Pro Phe Glu Arg
450 455 460
Asp Ile Ser Thr Glu Ile Tyr Gln Ala Gly Ser Lys Pro Cys Asn Gly
465 470 475 480
Val Glu Gly Phe Asn Cys Tyr Phe Pro Leu Gln Ser Tyr Gly Phe Gln
485 490 495
Pro Thr Asn Gly Val Gly Tyr Gln Pro Tyr Arg Val Val Val Leu Ser
500 505 510
Phe Glu Leu Leu His Ala Pro Ala Thr Val Cys Gly Pro Lys Lys Ser
515 520 525
Thr Asn Leu Val Lys Asn Lys Cys Val Asn Phe Asn Phe Asn Gly Leu
530 535 540
Thr Gly Thr Gly Val Leu Thr Glu Ser Asn Lys Lys Phe Leu Pro Phe
545 550 555 560
Gln Gln Phe Gly Arg Asp Ile Ala Asp Thr Thr Asp Ala Val Arg Asp
565 570 575
Pro Gln Thr Leu Glu Ile Leu Asp Ile Thr Pro Cys Ser Phe Gly Gly
580 585 590
Val Ser Val Ile Thr Pro Gly Thr Asn Thr Ser Asn Gln Val Ala Val
595 600 605
Leu Tyr Gln Gly Val Asn Cys Thr Glu Val Pro Val Ala Ile His Ala
610 615 620
Asp Gln Leu Thr Pro Thr Trp Arg Val Tyr Ser Thr Gly Ser Asn Val
625 630 635 640
Phe Gln Thr Arg Ala Gly Cys Leu Ile Gly Ala Glu His Val Asn Asn
645 650 655
Ser Tyr Glu Cys Asp Ile Pro Ile Gly Ala Gly Ile Cys Ala Ser Tyr
660 665 670
Gln Thr Gln Thr Asn Ser Arg Arg Arg Ala Arg Ser Val Ala Ser Gln
675 680 685
Ser Ile Ile Ala Tyr Thr Met Ser Leu Gly Ala Glu Asn Ser Val Ala
690 695 700
Tyr Ser Gln Asn Ser Ile Ala Ile Pro Thr Gln Phe Thr Ile Ser Val
705 710 715 720
Thr Thr Glu Ile Leu Pro Val Ser Met Thr Lys Thr Ser Val Asp Cys
725 730 735
Thr Met Tyr Ile Cys Gly Asp Ser Thr Glu Cys Ser Asn Leu Leu Leu
740 745 750
Gln Tyr Gly Ser Phe Cys Thr Gln Leu Asn Arg Ala Leu Thr Gly Ile
755 760 765
Ala Val Glu Gln Asp Lys Asn Thr Gln Glu Val Phe Ala Gln Val Lys
770 775 780
Gln Ile Tyr Lys Thr Pro Pro Ile Lys Asp Phe Gly Gly Phe Gln Phe
785 790 795 800
Ser Gln Ile Leu Pro Asp Pro Ser Lys Pro Ser Lys Arg Ser Phe Ile
805 810 815
Glu Asp Leu Leu Phe Asn Lys Val Thr Leu Ala Asp Ala Gly Phe Ile
820 825 830
Lys Gln Tyr Gly Asp Cys Leu Gly Asp Ile Ala Ala Arg Asp Leu Ile
835 840 845
Cys Ala Gln Lys Phe Asn Gly Leu Thr Val Leu Pro Pro Leu Leu Thr
850 855 860
Asp Glu Met Ile Ala Gln Tyr Thr Ser Ala Leu Leu Ala Gly Thr Ile
865 870 875 880
Thr Ser Gly Trp Thr Phe Gly Ala Gly Ala Ala Leu Gln Ile Pro Phe
885 890 895
Ala Met Gln Met Ala Tyr Arg Phe Asn Gly Ile Gly Val Thr Gln Asn
900 905 910
Val Leu Tyr Glu Asn Gln Lys Leu Ile Ala Asn Gln Phe Asn Ser Ala
915 920 925
Ile Gly Lys Ile Gln Asp Ser Leu Ser Ser Thr Ala Ser Ala Leu Gly
930 935 940
Lys Leu Gln Asn Val Val Asn Gln Asn Ala Gln Ala Leu Asn Thr Leu
945 950 955 960
Val Lys Gln Leu Ser Ser Asn Phe Gly Ala Ile Ser Ser Val Leu Asn
965 970 975
Asp Ile Leu Ser Arg Leu Asp Lys Val Glu Ala Glu Val Gln Ile Asp
980 985 990
Arg Leu Ile Thr Gly Arg Leu Gln Ser Leu Gln Thr Tyr Val Thr Gln
995 1000 1005
Gln Leu Ile Arg Ala Ala Glu Ile Arg Ala Ser Ala Asn Leu Ala
1010 1015 1020
Ala Thr Lys Met Ser Glu Cys Val Leu Gly Gln Ser Lys Arg Val
1025 1030 1035
Asp Phe Cys Gly Lys Gly Tyr His Leu Met Ser Phe Pro Gln Ser
1040 1045 1050
Ala Pro His Gly Val Val Phe Leu His Val Thr Tyr Val Pro Ala
1055 1060 1065
Gln Glu Lys Gln Phe Thr Thr Ala Pro Ala Ile Cys His Asp Gly
1070 1075 1080
Lys Ala His Phe Pro Arg Glu Gly Val Phe Val Ser Gln Gly Thr
1085 1090 1095
His Trp Phe Val Thr Gln Arg Asn Phe Tyr Glu Pro Gln Ile Ile
1100 1105 1110
Thr Thr Asp Asn Thr Phe Val Ser Gly Asn Cys Asp Val Val Ile
1115 1120 1125
Gly Ile Val Gln Asn Thr Val Tyr Asp Pro Leu Gln Pro Glu Leu
1130 1135 1140
Asp Ser Phe Lys Glu Glu Leu Asp Lys Tyr Phe Lys Gln His Thr
1145 1150 1155
Ser Pro Asp Val Asp Leu Gly Asp Ile Ser Gly Ile Gln Ala Ser
1160 1165 1170
Val Val Asn Ile Gln Lys Glu Ile Asp Arg Leu Asn Glu Val Ala
1175 1180 1185
Lys Asn Leu Gln Glu Ser Leu Ile Asp Leu Gln Glu Leu Gly Lys
1190 1195 1200
Tyr Glu Gln Tyr Ile Lys Trp Pro Trp Tyr Ile Trp Leu Gly Phe
1205 1210 1215
Ile Ala Gly Leu Ile Ala Ile Val Met Val Thr Ile Met Leu Cys
1220 1225 1230
Cys Met Thr Ser Cys Cys Ser Cys Leu Lys Gly Cys Cys Ser Cys
1235 1240 1245
Gly Ser Cys Cys Lys Phe Asp Glu Asp Asp Ser Glu Pro Val Leu
1250 1255 1260
Lys Gly Val Lys Leu His Tyr Thr
1265 1270
<210> 29
<211> 3822
<212> DNA
<213> Artificial Sequence
<220>
<223> S-(deg-S2) for Wuhan S-2P
<400> 29
atgttcgtct tcctggtcct gctgcctctg gtctcctcac agtgcgtcaa tctgacaact 60
cggactcagc tgccacctgc ttatactaat agcttcacca gaggcgtgta ctatcctgac 120
aaggtgttta gaagctccgt gctgcactct acacaggatc tgtttctgcc attctttagc 180
aacgtgacct ggttccacgc catccacgtg agcggcacca atggcacaaa gcggttcgac 240
aatcccgtgc tgccttttaa cgatggcgtg tacttcgcct ctaccgagaa gagcaacatc 300
atcagaggct ggatctttgg caccacactg gactccaaga cacagtctct gctgatcgtg 360
aacaatgcca ccaacgtggt catcaaggtg tgcgagttcc agttttgtaa tgatcccttc 420
ctgggcgtgt actatcacaa gaacaataag agctggatgg agtccgagtt tagagtgtat 480
tctagcgcca acaactgcac atttgagtac gtgagccagc ctttcctgat ggacctggag 540
ggcaagcagg gcaatttcaa gaacctgagg gagttcgtgt ttaagaatat cgacggctac 600
ttcaaaatct actctaagca cacccccatc aacctggtgc gcgacctgcc tcagggcttc 660
agcgccctgg agcccctggt ggatctgcct atcggcatca acatcacccg gtttcagaca 720
ctgctggccc tgcacagaag ctacctgaca cccggcgact cctctagcgg atggaccgcc 780
ggcgctgccg cctactatgt gggctacctc cagccccgga ccttcctgct gaagtacaac 840
gagaatggca ccatcacaga cgcagtggat tgcgccctgg accccctgag cgagacaaag 900
tgtacactga agtcctttac cgtggagaag ggcatctatc agacatccaa tttcagggtg 960
cagccaaccg agtctatcgt gcgctttcct aatatcacaa acctgtgccc atttggcgag 1020
gtgttcaacg caacccgctt cgccagcgtg tacgcctgga ataggaagcg gatcagcaac 1080
tgcgtggccg actatagcgt gctgtacaac tccgcctctt tcagcacctt taagtgctat 1140
ggcgtgtccc ccacaaagct gaatgacctg tgctttacca acgtctacgc cgattctttc 1200
gtgatcaggg gcgacgaggt gcgccagatc gcccccggcc agacaggcaa gatcgcagac 1260
tacaattata agctgccaga cgatttcacc ggctgcgtga tcgcctggaa cagcaacaat 1320
ctggattcca aagtgggcgg caactacaat tatctgtacc ggctgtttag aaagagcaat 1380
ctgaagccct tcgagaggga catctctaca gaaatctacc aggccggcag caccccttgc 1440
aatggcgtgg agggctttaa ctgttatttc ccactccagt cctacggctt ccagcccaca 1500
aacggcgtgg gctatcagcc ttaccgcgtg gtggtgctga gctttgagct gctgcacgcc 1560
ccagcaacag tgtgcggccc caagaagtcc accaatctgg tgaagaacaa gtgcgtgaac 1620
ttcaacttca acggcctgac cggcacaggc gtgctgaccg agtccaacaa gaagttcctg 1680
ccatttcagc agttcggcag ggacatcgca gataccacag acgccgtgcg cgacccacag 1740
accctggaga tcctggacat cacaccctgc tctttcggcg gcgtgagcgt gatcacaccc 1800
ggcaccaata caagcaacca ggtggccgtg ctgtatcagg acgtgaattg taccgaggtg 1860
cccgtggcta tccacgccga tcagctgacc ccaacatggc gggtgtacag caccggctcc 1920
aacgtcttcc agacaagagc cggatgcctg atcggagcag agcacgtgaa caattcctat 1980
gagtgcgaca tcccaatcgg cgccggcatc tgtgcctctt accagaccca gacaaactct 2040
cccagaagag cccggagcgt ggcctcccag tctatcatcg cctataccat gtccctgggc 2100
gccgagaaca gcgtggccta ctctcagaat agcatcgcca tcccaaccca gttcacaatc 2160
tctgtgacca cagagatcct gcccgtgtcc atgaccaaga catctgtgga ctgcacaatg 2220
tatatctgtg gcgattctac cgagtgcagc aacctgctgc tccagtacgg cagcttttgt 2280
acccagctga atagagccct gacaggcatc gccgtggagc aggataagaa cacacaggag 2340
gtgttcgccc aggtgaagca aatctacaag acccccccta tcaaggactt tggcggcttc 2400
caattttccc agatcctgcc tgatccatcc aagccttcta agcggagctt tatcgaggac 2460
ctgctgttca acaaggtgac cctggccgat gccggcttca tcaagcagta tggcgattgc 2520
ctgggcgaca tcgcagccag ggacctgatc tgcgcccaga agtttaatgg cctgaccgtg 2580
ctgccacccc tgctgacaga tgagatgatc gcacagtaca caagcgccct gctggccggc 2640
accatcacat ccggatggac cttcggcgca ggagccgccc tccagatccc ctttgccatg 2700
cagatggcct ataggttcaa cggcatcggc gtgacccaga atgtgctgta cgagaaccag 2760
aagctgatcg ccaatcagtt taactccgcc atcggcaaga tccaggacag cctgtcctct 2820
acagccagcg ccctgggcaa gctccaggat gtggtgaatc agaacgccca ggccctgaat 2880
accctggtga agcagctgag cagcaacttc ggcgccatct ctagcgtgct gaatgacatc 2940
ctgagccggc tggacccgcc ggaggcagag gtgcagatcg accggctgat caccggccgg 3000
ctccagagcc tccagaccta tgtgacacag cagctgatca gggccgccga gatcagggcc 3060
agcgccaatc tggcagcaac caagatgtcc gagtgcgtgc tgggccagtc taagagagtg 3120
gacttttgtg gcaagggcta tcacctgatg tccttccctc agtctgcccc acacggcgtg 3180
gtgtttctgc acgtgaccta cgtgcccgcc caggagaagc agttcaccac agcccctgcc 3240
atctgccacg atggcaaggc ccactttcca agggagggcg tgttcgtgtc ccagggcacc 3300
cactggtttg tgacacagcg caatttctac gagccccaga tcatcaccac agacaacacc 3360
ttcgtgagcg gcaactgtga cgtggtcatc ggcatcgtgc agaataccgt gtatgatcca 3420
ctccagcccg agctggacag ctttaaggag gagctggata agtatttcaa gcaacacacc 3480
tcccctgacg tggatctggg cgacatcagc ggcatccaag cctccgtggt gaacatccag 3540
aaggagatcg accgcctgaa cgaggtggct aagaatctgc aggagagcct gatcgacctc 3600
caggagctgg gcaagtatga gcagtacatc aagtggccct ggtacatctg gctgggcttc 3660
atcgccggcc tgatcgccat cgtgatggtg accatcatgc tgtgctgtat gacatcctgc 3720
tgttcttgcc tgaagggctg ctgtagctgt ggctcctgct gtaagtttga cgaggatgac 3780
tctgaacctg tgctgaaggg cgtgaagctg cattacacct aa 3822
<210> 30
<211> 1273
<212> PRT
<213> Artificial Sequence
<220>
<223> S-(deg-S2) for Wuhan S-2P
<400> 30
Met Phe Val Phe Leu Val Leu Leu Pro Leu Val Ser Ser Gln Cys Val
1 5 10 15
Asn Leu Thr Thr Arg Thr Gln Leu Pro Pro Ala Tyr Thr Asn Ser Phe
20 25 30
Thr Arg Gly Val Tyr Tyr Pro Asp Lys Val Phe Arg Ser Ser Val Leu
35 40 45
His Ser Thr Gln Asp Leu Phe Leu Pro Phe Phe Ser Asn Val Thr Trp
50 55 60
Phe His Ala Ile His Val Ser Gly Thr Asn Gly Thr Lys Arg Phe Asp
65 70 75 80
Asn Pro Val Leu Pro Phe Asn Asp Gly Val Tyr Phe Ala Ser Thr Glu
85 90 95
Lys Ser Asn Ile Ile Arg Gly Trp Ile Phe Gly Thr Thr Leu Asp Ser
100 105 110
Lys Thr Gln Ser Leu Leu Ile Val Asn Asn Ala Thr Asn Val Val Ile
115 120 125
Lys Val Cys Glu Phe Gln Phe Cys Asn Asp Pro Phe Leu Gly Val Tyr
130 135 140
Tyr His Lys Asn Asn Lys Ser Trp Met Glu Ser Glu Phe Arg Val Tyr
145 150 155 160
Ser Ser Ala Asn Asn Cys Thr Phe Glu Tyr Val Ser Gln Pro Phe Leu
165 170 175
Met Asp Leu Glu Gly Lys Gln Gly Asn Phe Lys Asn Leu Arg Glu Phe
180 185 190
Val Phe Lys Asn Ile Asp Gly Tyr Phe Lys Ile Tyr Ser Lys His Thr
195 200 205
Pro Ile Asn Leu Val Arg Asp Leu Pro Gln Gly Phe Ser Ala Leu Glu
210 215 220
Pro Leu Val Asp Leu Pro Ile Gly Ile Asn Ile Thr Arg Phe Gln Thr
225 230 235 240
Leu Leu Ala Leu His Arg Ser Tyr Leu Thr Pro Gly Asp Ser Ser Ser
245 250 255
Gly Trp Thr Ala Gly Ala Ala Ala Tyr Tyr Val Gly Tyr Leu Gln Pro
260 265 270
Arg Thr Phe Leu Leu Lys Tyr Asn Glu Asn Gly Thr Ile Thr Asp Ala
275 280 285
Val Asp Cys Ala Leu Asp Pro Leu Ser Glu Thr Lys Cys Thr Leu Lys
290 295 300
Ser Phe Thr Val Glu Lys Gly Ile Tyr Gln Thr Ser Asn Phe Arg Val
305 310 315 320
Gln Pro Thr Glu Ser Ile Val Arg Phe Pro Asn Ile Thr Asn Leu Cys
325 330 335
Pro Phe Gly Glu Val Phe Asn Ala Thr Arg Phe Ala Ser Val Tyr Ala
340 345 350
Trp Asn Arg Lys Arg Ile Ser Asn Cys Val Ala Asp Tyr Ser Val Leu
355 360 365
Tyr Asn Ser Ala Ser Phe Ser Thr Phe Lys Cys Tyr Gly Val Ser Pro
370 375 380
Thr Lys Leu Asn Asp Leu Cys Phe Thr Asn Val Tyr Ala Asp Ser Phe
385 390 395 400
Val Ile Arg Gly Asp Glu Val Arg Gln Ile Ala Pro Gly Gln Thr Gly
405 410 415
Lys Ile Ala Asp Tyr Asn Tyr Lys Leu Pro Asp Asp Phe Thr Gly Cys
420 425 430
Val Ile Ala Trp Asn Ser Asn Asn Leu Asp Ser Lys Val Gly Gly Asn
435 440 445
Tyr Asn Tyr Leu Tyr Arg Leu Phe Arg Lys Ser Asn Leu Lys Pro Phe
450 455 460
Glu Arg Asp Ile Ser Thr Glu Ile Tyr Gln Ala Gly Ser Thr Pro Cys
465 470 475 480
Asn Gly Val Glu Gly Phe Asn Cys Tyr Phe Pro Leu Gln Ser Tyr Gly
485 490 495
Phe Gln Pro Thr Asn Gly Val Gly Tyr Gln Pro Tyr Arg Val Val Val
500 505 510
Leu Ser Phe Glu Leu Leu His Ala Pro Ala Thr Val Cys Gly Pro Lys
515 520 525
Lys Ser Thr Asn Leu Val Lys Asn Lys Cys Val Asn Phe Asn Phe Asn
530 535 540
Gly Leu Thr Gly Thr Gly Val Leu Thr Glu Ser Asn Lys Lys Phe Leu
545 550 555 560
Pro Phe Gln Gln Phe Gly Arg Asp Ile Ala Asp Thr Thr Asp Ala Val
565 570 575
Arg Asp Pro Gln Thr Leu Glu Ile Leu Asp Ile Thr Pro Cys Ser Phe
580 585 590
Gly Gly Val Ser Val Ile Thr Pro Gly Thr Asn Thr Ser Asn Gln Val
595 600 605
Ala Val Leu Tyr Gln Asp Val Asn Cys Thr Glu Val Pro Val Ala Ile
610 615 620
His Ala Asp Gln Leu Thr Pro Thr Trp Arg Val Tyr Ser Thr Gly Ser
625 630 635 640
Asn Val Phe Gln Thr Arg Ala Gly Cys Leu Ile Gly Ala Glu His Val
645 650 655
Asn Asn Ser Tyr Glu Cys Asp Ile Pro Ile Gly Ala Gly Ile Cys Ala
660 665 670
Ser Tyr Gln Thr Gln Thr Asn Ser Pro Arg Arg Ala Arg Ser Val Ala
675 680 685
Ser Gln Ser Ile Ile Ala Tyr Thr Met Ser Leu Gly Ala Glu Asn Ser
690 695 700
Val Ala Tyr Ser Gln Asn Ser Ile Ala Ile Pro Thr Gln Phe Thr Ile
705 710 715 720
Ser Val Thr Thr Glu Ile Leu Pro Val Ser Met Thr Lys Thr Ser Val
725 730 735
Asp Cys Thr Met Tyr Ile Cys Gly Asp Ser Thr Glu Cys Ser Asn Leu
740 745 750
Leu Leu Gln Tyr Gly Ser Phe Cys Thr Gln Leu Asn Arg Ala Leu Thr
755 760 765
Gly Ile Ala Val Glu Gln Asp Lys Asn Thr Gln Glu Val Phe Ala Gln
770 775 780
Val Lys Gln Ile Tyr Lys Thr Pro Pro Ile Lys Asp Phe Gly Gly Phe
785 790 795 800
Gln Phe Ser Gln Ile Leu Pro Asp Pro Ser Lys Pro Ser Lys Arg Ser
805 810 815
Phe Ile Glu Asp Leu Leu Phe Asn Lys Val Thr Leu Ala Asp Ala Gly
820 825 830
Phe Ile Lys Gln Tyr Gly Asp Cys Leu Gly Asp Ile Ala Ala Arg Asp
835 840 845
Leu Ile Cys Ala Gln Lys Phe Asn Gly Leu Thr Val Leu Pro Pro Leu
850 855 860
Leu Thr Asp Glu Met Ile Ala Gln Tyr Thr Ser Ala Leu Leu Ala Gly
865 870 875 880
Thr Ile Thr Ser Gly Trp Thr Phe Gly Ala Gly Ala Ala Leu Gln Ile
885 890 895
Pro Phe Ala Met Gln Met Ala Tyr Arg Phe Asn Gly Ile Gly Val Thr
900 905 910
Gln Asn Val Leu Tyr Glu Asn Gln Lys Leu Ile Ala Asn Gln Phe Asn
915 920 925
Ser Ala Ile Gly Lys Ile Gln Asp Ser Leu Ser Ser Thr Ala Ser Ala
930 935 940
Leu Gly Lys Leu Gln Asp Val Val Asn Gln Asn Ala Gln Ala Leu Asn
945 950 955 960
Thr Leu Val Lys Gln Leu Ser Ser Asn Phe Gly Ala Ile Ser Ser Val
965 970 975
Leu Asn Asp Ile Leu Ser Arg Leu Asp Pro Pro Glu Ala Glu Val Gln
980 985 990
Ile Asp Arg Leu Ile Thr Gly Arg Leu Gln Ser Leu Gln Thr Tyr Val
995 1000 1005
Thr Gln Gln Leu Ile Arg Ala Ala Glu Ile Arg Ala Ser Ala Asn
1010 1015 1020
Leu Ala Ala Thr Lys Met Ser Glu Cys Val Leu Gly Gln Ser Lys
1025 1030 1035
Arg Val Asp Phe Cys Gly Lys Gly Tyr His Leu Met Ser Phe Pro
1040 1045 1050
Gln Ser Ala Pro His Gly Val Val Phe Leu His Val Thr Tyr Val
1055 1060 1065
Pro Ala Gln Glu Lys Gln Phe Thr Thr Ala Pro Ala Ile Cys His
1070 1075 1080
Asp Gly Lys Ala His Phe Pro Arg Glu Gly Val Phe Val Ser Gln
1085 1090 1095
Gly Thr His Trp Phe Val Thr Gln Arg Asn Phe Tyr Glu Pro Gln
1100 1105 1110
Ile Ile Thr Thr Asp Asn Thr Phe Val Ser Gly Asn Cys Asp Val
1115 1120 1125
Val Ile Gly Ile Val Gln Asn Thr Val Tyr Asp Pro Leu Gln Pro
1130 1135 1140
Glu Leu Asp Ser Phe Lys Glu Glu Leu Asp Lys Tyr Phe Lys Gln
1145 1150 1155
His Thr Ser Pro Asp Val Asp Leu Gly Asp Ile Ser Gly Ile Gln
1160 1165 1170
Ala Ser Val Val Asn Ile Gln Lys Glu Ile Asp Arg Leu Asn Glu
1175 1180 1185
Val Ala Lys Asn Leu Gln Glu Ser Leu Ile Asp Leu Gln Glu Leu
1190 1195 1200
Gly Lys Tyr Glu Gln Tyr Ile Lys Trp Pro Trp Tyr Ile Trp Leu
1205 1210 1215
Gly Phe Ile Ala Gly Leu Ile Ala Ile Val Met Val Thr Ile Met
1220 1225 1230
Leu Cys Cys Met Thr Ser Cys Cys Ser Cys Leu Lys Gly Cys Cys
1235 1240 1245
Ser Cys Gly Ser Cys Cys Lys Phe Asp Glu Asp Asp Ser Glu Pro
1250 1255 1260
Val Leu Lys Gly Val Lys Leu His Tyr Thr
1265 1270
<210> 31
<211> 3816
<212> DNA
<213> Artificial Sequence
<220>
<223> S-(deg-S2) for Delta S-2P strain
<400> 31
atgttcgtct tcctggtcct gctgcctctg gtctcctcac agtgcgtcaa tctgcgaact 60
cggactcagc tgccacctgc ttatactaat agcttcacca gaggcgtgta ctatcctgac 120
aaggtgttta gaagctccgt gctgcactct acacaggatc tgtttctgcc attctttagc 180
aacgtgacct ggttccacgc catccacgtg agcggcacca atggcacaaa gcggttcgac 240
aatcccgtgc tgccttttaa cgatggcgtg tacttcgcct ctatcgagaa gagcaacatc 300
atcagaggct ggatctttgg caccacactg gactccaaga cacagtctct gctgatcgtg 360
aacaatgcca ccaacgtggt catcaaggtg tgcgagttcc agttttgtaa tgatcccttc 420
ctggacgtgt actatcacaa gaacaataag agctggatgg agtccggagt gtattctagc 480
gccaacaact gcacatttga gtacgtgagc cagcctttcc tgatggacct ggagggcaag 540
cagggcaatt tcaagaacct gagggagttc gtgtttaaga atatcgacgg ctacttcaaa 600
atctactcta agcacacccc catcaacctg gtgcgcgacc tgcctcaggg cttcagcgcc 660
ctggagcccc tggtggatct gcctatcggc atcaacatca cccggtttca gacactgctg 720
gccctgcaca gaagctacct gacacccggc gactcctcta gcggatggac cgccggcgct 780
gccgcctact atgtgggcta cctccagccc cggaccttcc tgctgaagta caacgagaat 840
ggcaccatca cagacgcagt ggattgcgcc ctggaccccc tgagcgagac aaagtgtaca 900
ctgaagtcct ttaccgtgga gaagggcatc tatcagacat ccaatttcag ggtgcagcca 960
accgagtcta tcgtgcgctt tcctaatatc acaaacctgt gcccatttgg cgaggtgttc 1020
aacgcaaccc gcttcgccag cgtgtacgcc tggaatagga agcggatcag caactgcgtg 1080
gccgactata gcgtgctgta caactccgcc tctttcagca cctttaagtg ctatggcgtg 1140
tcccccacaa agctgaatga cctgtgcttt accaacgtct acgccgattc tttcgtgatc 1200
aggggcgacg aggtgcgcca gatcgccccc ggccagacag gcaagatcgc agactacaat 1260
tataagctgc cagacgattt caccggctgc gtgatcgcct ggaacagcaa caatctggat 1320
tccaaagtgg gcggcaacta caattatcgg taccggctgt ttagaaagag caatctgaag 1380
cccttcgaga gggacatctc tacagaaatc taccaggccg gcagcaagcc ttgcaatggc 1440
gtggagggct ttaactgtta tttcccactc cagtcctacg gcttccagcc cacaaacggc 1500
gtgggctatc agccttaccg cgtggtggtg ctgagctttg agctgctgca cgccccagca 1560
acagtgtgcg gccccaagaa gtccaccaat ctggtgaaga acaagtgcgt gaacttcaac 1620
ttcaacggcc tgaccggcac aggcgtgctg accgagtcca acaagaagtt cctgccattt 1680
cagcagttcg gcagggacat cgcagatacc acagacgccg tgcgcgaccc acagaccctg 1740
gagatcctgg acatcacacc ctgctctttc ggcggcgtga gcgtgatcac acccggcacc 1800
aatacaagca accaggtggc cgtgctgtat cagggcgtga attgtaccga ggtgcccgtg 1860
gctatccacg ccgatcagct gaccccaaca tggcgggtgt acagcaccgg ctccaacgtc 1920
ttccagacaa gagccggatg cctgatcgga gcagagcacg tgaacaattc ctatgagtgc 1980
gacatcccaa tcggcgccgg catctgtgcc tcttaccaga cccagacaaa ctctcgcaga 2040
agagcccgga gcgtggcctc ccagtctatc atcgcctata ccatgtccct gggcgccgag 2100
aacagcgtgg cctactctca gaatagcatc gccatcccaa cccagttcac aatctctgtg 2160
accacagaga tcctgcccgt gtccatgacc aagacatctg tggactgcac aatgtatatc 2220
tgtggcgatt ctaccgagtg cagcaacctg ctgctccagt acggcagctt ttgtacccag 2280
ctgaatagag ccctgacagg catcgccgtg gagcaggata agaacacaca ggaggtgttc 2340
gcccaggtga agcaaatcta caagaccccc cctatcaagg actttggcgg cttccaattt 2400
tcccagatcc tgcctgatcc atccaagcct tctaagcgga gctttatcga ggacctgctg 2460
ttcaacaagg tgaccctggc cgatgccggc ttcatcaagc agtatggcga ttgcctgggc 2520
gacatcgcag ccagggacct gatctgcgcc cagaagttta atggcctgac cgtgctgcca 2580
cccctgctga cagatgagat gatcgcacag tacacaagcg ccctgctggc cggcaccatc 2640
acatccggat ggaccttcgg cgcaggagcc gccctccaga tcccctttgc catgcagatg 2700
gcctataggt tcaacggcat cggcgtgacc cagaatgtgc tgtacgagaa ccagaagctg 2760
atcgccaatc agtttaactc cgccatcggc aagatccagg acagcctgtc ctctacagcc 2820
agcgccctgg gcaagctcca gaatgtggtg aatcagaacg cccaggccct gaataccctg 2880
gtgaagcagc tgagcagcaa cttcggcgcc atctctagcg tgctgaatga catcctgagc 2940
cggctggacc gccgggaggc agaggtgcag atcgaccggc tgatcaccgg ccggctccag 3000
agcctccaga cctatgtgac acagcagctg atcagggccg ccgagatcag ggccagcgcc 3060
aatctggcag caaccaagat gtccgagtgc gtgctgggcc agtctaagag agtggacttt 3120
tgtggcaagg gctatcacct gatgtccttc cctcagtctg ccccacacgg cgtggtgttt 3180
ctgcacgtga cctacgtgcc cgcccaggag aagcagttca ccacagcccc tgccatctgc 3240
cacgatggca aggcccactt tccaagggag ggcgtgttcg tgtcccaggg cacccactgg 3300
tttgtgacac agcgcaattt ctacgagccc cagatcatca ccacagacaa caccttcgtg 3360
agcggcaact gtgacgtggt catcggcatc gtgcagaata ccgtgtatga tccactccag 3420
cccgagctgg acagctttaa ggaggagctg gataagtatt tcaagcaaca cacctcccct 3480
gacgtggatc tgggcgacat cagcggcatc caagcctccg tggtgaacat ccagaaggag 3540
atcgaccgcc tgaacgaggt ggctaagaat ctgcaggaga gcctgatcga cctccaggag 3600
ctgggcaagt atgagcagta catcaagtgg ccctggtaca tctggctggg cttcatcgcc 3660
ggcctgatcg ccatcgtgat ggtgaccatc atgctgtgct gtatgacatc ctgctgttct 3720
tgcctgaagg gctgctgtag ctgtggctcc tgctgtaagt ttgacgagga tgactctgaa 3780
cctgtgctga agggcgtgaa gctgcattac acctaa 3816
<210> 32
<211> 1271
<212> PRT
<213> Artificial Sequence
<220>
<223> S-(deg-S2) for Delta S-2P strain
<400> 32
Met Phe Val Phe Leu Val Leu Leu Pro Leu Val Ser Ser Gln Cys Val
1 5 10 15
Asn Leu Arg Thr Arg Thr Gln Leu Pro Pro Ala Tyr Thr Asn Ser Phe
20 25 30
Thr Arg Gly Val Tyr Tyr Pro Asp Lys Val Phe Arg Ser Ser Val Leu
35 40 45
His Ser Thr Gln Asp Leu Phe Leu Pro Phe Phe Ser Asn Val Thr Trp
50 55 60
Phe His Ala Ile His Val Ser Gly Thr Asn Gly Thr Lys Arg Phe Asp
65 70 75 80
Asn Pro Val Leu Pro Phe Asn Asp Gly Val Tyr Phe Ala Ser Ile Glu
85 90 95
Lys Ser Asn Ile Ile Arg Gly Trp Ile Phe Gly Thr Thr Leu Asp Ser
100 105 110
Lys Thr Gln Ser Leu Leu Ile Val Asn Asn Ala Thr Asn Val Val Ile
115 120 125
Lys Val Cys Glu Phe Gln Phe Cys Asn Asp Pro Phe Leu Asp Val Tyr
130 135 140
Tyr His Lys Asn Asn Lys Ser Trp Met Glu Ser Gly Val Tyr Ser Ser
145 150 155 160
Ala Asn Asn Cys Thr Phe Glu Tyr Val Ser Gln Pro Phe Leu Met Asp
165 170 175
Leu Glu Gly Lys Gln Gly Asn Phe Lys Asn Leu Arg Glu Phe Val Phe
180 185 190
Lys Asn Ile Asp Gly Tyr Phe Lys Ile Tyr Ser Lys His Thr Pro Ile
195 200 205
Asn Leu Val Arg Asp Leu Pro Gln Gly Phe Ser Ala Leu Glu Pro Leu
210 215 220
Val Asp Leu Pro Ile Gly Ile Asn Ile Thr Arg Phe Gln Thr Leu Leu
225 230 235 240
Ala Leu His Arg Ser Tyr Leu Thr Pro Gly Asp Ser Ser Ser Gly Trp
245 250 255
Thr Ala Gly Ala Ala Ala Tyr Tyr Val Gly Tyr Leu Gln Pro Arg Thr
260 265 270
Phe Leu Leu Lys Tyr Asn Glu Asn Gly Thr Ile Thr Asp Ala Val Asp
275 280 285
Cys Ala Leu Asp Pro Leu Ser Glu Thr Lys Cys Thr Leu Lys Ser Phe
290 295 300
Thr Val Glu Lys Gly Ile Tyr Gln Thr Ser Asn Phe Arg Val Gln Pro
305 310 315 320
Thr Glu Ser Ile Val Arg Phe Pro Asn Ile Thr Asn Leu Cys Pro Phe
325 330 335
Gly Glu Val Phe Asn Ala Thr Arg Phe Ala Ser Val Tyr Ala Trp Asn
340 345 350
Arg Lys Arg Ile Ser Asn Cys Val Ala Asp Tyr Ser Val Leu Tyr Asn
355 360 365
Ser Ala Ser Phe Ser Thr Phe Lys Cys Tyr Gly Val Ser Pro Thr Lys
370 375 380
Leu Asn Asp Leu Cys Phe Thr Asn Val Tyr Ala Asp Ser Phe Val Ile
385 390 395 400
Arg Gly Asp Glu Val Arg Gln Ile Ala Pro Gly Gln Thr Gly Lys Ile
405 410 415
Ala Asp Tyr Asn Tyr Lys Leu Pro Asp Asp Phe Thr Gly Cys Val Ile
420 425 430
Ala Trp Asn Ser Asn Asn Leu Asp Ser Lys Val Gly Gly Asn Tyr Asn
435 440 445
Tyr Arg Tyr Arg Leu Phe Arg Lys Ser Asn Leu Lys Pro Phe Glu Arg
450 455 460
Asp Ile Ser Thr Glu Ile Tyr Gln Ala Gly Ser Lys Pro Cys Asn Gly
465 470 475 480
Val Glu Gly Phe Asn Cys Tyr Phe Pro Leu Gln Ser Tyr Gly Phe Gln
485 490 495
Pro Thr Asn Gly Val Gly Tyr Gln Pro Tyr Arg Val Val Val Leu Ser
500 505 510
Phe Glu Leu Leu His Ala Pro Ala Thr Val Cys Gly Pro Lys Lys Ser
515 520 525
Thr Asn Leu Val Lys Asn Lys Cys Val Asn Phe Asn Phe Asn Gly Leu
530 535 540
Thr Gly Thr Gly Val Leu Thr Glu Ser Asn Lys Lys Phe Leu Pro Phe
545 550 555 560
Gln Gln Phe Gly Arg Asp Ile Ala Asp Thr Thr Asp Ala Val Arg Asp
565 570 575
Pro Gln Thr Leu Glu Ile Leu Asp Ile Thr Pro Cys Ser Phe Gly Gly
580 585 590
Val Ser Val Ile Thr Pro Gly Thr Asn Thr Ser Asn Gln Val Ala Val
595 600 605
Leu Tyr Gln Gly Val Asn Cys Thr Glu Val Pro Val Ala Ile His Ala
610 615 620
Asp Gln Leu Thr Pro Thr Trp Arg Val Tyr Ser Thr Gly Ser Asn Val
625 630 635 640
Phe Gln Thr Arg Ala Gly Cys Leu Ile Gly Ala Glu His Val Asn Asn
645 650 655
Ser Tyr Glu Cys Asp Ile Pro Ile Gly Ala Gly Ile Cys Ala Ser Tyr
660 665 670
Gln Thr Gln Thr Asn Ser Arg Arg Arg Ala Arg Ser Val Ala Ser Gln
675 680 685
Ser Ile Ile Ala Tyr Thr Met Ser Leu Gly Ala Glu Asn Ser Val Ala
690 695 700
Tyr Ser Gln Asn Ser Ile Ala Ile Pro Thr Gln Phe Thr Ile Ser Val
705 710 715 720
Thr Thr Glu Ile Leu Pro Val Ser Met Thr Lys Thr Ser Val Asp Cys
725 730 735
Thr Met Tyr Ile Cys Gly Asp Ser Thr Glu Cys Ser Asn Leu Leu Leu
740 745 750
Gln Tyr Gly Ser Phe Cys Thr Gln Leu Asn Arg Ala Leu Thr Gly Ile
755 760 765
Ala Val Glu Gln Asp Lys Asn Thr Gln Glu Val Phe Ala Gln Val Lys
770 775 780
Gln Ile Tyr Lys Thr Pro Pro Ile Lys Asp Phe Gly Gly Phe Gln Phe
785 790 795 800
Ser Gln Ile Leu Pro Asp Pro Ser Lys Pro Ser Lys Arg Ser Phe Ile
805 810 815
Glu Asp Leu Leu Phe Asn Lys Val Thr Leu Ala Asp Ala Gly Phe Ile
820 825 830
Lys Gln Tyr Gly Asp Cys Leu Gly Asp Ile Ala Ala Arg Asp Leu Ile
835 840 845
Cys Ala Gln Lys Phe Asn Gly Leu Thr Val Leu Pro Pro Leu Leu Thr
850 855 860
Asp Glu Met Ile Ala Gln Tyr Thr Ser Ala Leu Leu Ala Gly Thr Ile
865 870 875 880
Thr Ser Gly Trp Thr Phe Gly Ala Gly Ala Ala Leu Gln Ile Pro Phe
885 890 895
Ala Met Gln Met Ala Tyr Arg Phe Asn Gly Ile Gly Val Thr Gln Asn
900 905 910
Val Leu Tyr Glu Asn Gln Lys Leu Ile Ala Asn Gln Phe Asn Ser Ala
915 920 925
Ile Gly Lys Ile Gln Asp Ser Leu Ser Ser Thr Ala Ser Ala Leu Gly
930 935 940
Lys Leu Gln Asn Val Val Asn Gln Asn Ala Gln Ala Leu Asn Thr Leu
945 950 955 960
Val Lys Gln Leu Ser Ser Asn Phe Gly Ala Ile Ser Ser Val Leu Asn
965 970 975
Asp Ile Leu Ser Arg Leu Asp Pro Pro Glu Ala Glu Val Gln Ile Asp
980 985 990
Arg Leu Ile Thr Gly Arg Leu Gln Ser Leu Gln Thr Tyr Val Thr Gln
995 1000 1005
Gln Leu Ile Arg Ala Ala Glu Ile Arg Ala Ser Ala Asn Leu Ala
1010 1015 1020
Ala Thr Lys Met Ser Glu Cys Val Leu Gly Gln Ser Lys Arg Val
1025 1030 1035
Asp Phe Cys Gly Lys Gly Tyr His Leu Met Ser Phe Pro Gln Ser
1040 1045 1050
Ala Pro His Gly Val Val Phe Leu His Val Thr Tyr Val Pro Ala
1055 1060 1065
Gln Glu Lys Gln Phe Thr Thr Ala Pro Ala Ile Cys His Asp Gly
1070 1075 1080
Lys Ala His Phe Pro Arg Glu Gly Val Phe Val Ser Gln Gly Thr
1085 1090 1095
His Trp Phe Val Thr Gln Arg Asn Phe Tyr Glu Pro Gln Ile Ile
1100 1105 1110
Thr Thr Asp Asn Thr Phe Val Ser Gly Asn Cys Asp Val Val Ile
1115 1120 1125
Gly Ile Val Gln Asn Thr Val Tyr Asp Pro Leu Gln Pro Glu Leu
1130 1135 1140
Asp Ser Phe Lys Glu Glu Leu Asp Lys Tyr Phe Lys Gln His Thr
1145 1150 1155
Ser Pro Asp Val Asp Leu Gly Asp Ile Ser Gly Ile Gln Ala Ser
1160 1165 1170
Val Val Asn Ile Gln Lys Glu Ile Asp Arg Leu Asn Glu Val Ala
1175 1180 1185
Lys Asn Leu Gln Glu Ser Leu Ile Asp Leu Gln Glu Leu Gly Lys
1190 1195 1200
Tyr Glu Gln Tyr Ile Lys Trp Pro Trp Tyr Ile Trp Leu Gly Phe
1205 1210 1215
Ile Ala Gly Leu Ile Ala Ile Val Met Val Thr Ile Met Leu Cys
1220 1225 1230
Cys Met Thr Ser Cys Cys Ser Cys Leu Lys Gly Cys Cys Ser Cys
1235 1240 1245
Gly Ser Cys Cys Lys Phe Asp Glu Asp Asp Ser Glu Pro Val Leu
1250 1255 1260
Lys Gly Val Lys Leu His Tyr Thr
1265 1270
<210> 33
<211> 3822
<212> DNA
<213> Artificial Sequence
<220>
<223> S-(S2-1194) for Wuhan S-2P strain
<400> 33
atgttcgtct tcctggtcct gctgcctctg gtctcctcac agtgcgtcaa tctgacaact 60
cggactcagc tgccacctgc ttatactaat agcttcacca gaggcgtgta ctatcctgac 120
aaggtgttta gaagctccgt gctgcactct acacaggatc tgtttctgcc attctttagc 180
aacgtgacct ggttccacgc catccacgtg agcggcacca atggcacaaa gcggttcgac 240
aatcccgtgc tgccttttaa cgatggcgtg tacttcgcct ctaccgagaa gagcaacatc 300
atcagaggct ggatctttgg caccacactg gactccaaga cacagtctct gctgatcgtg 360
aacaatgcca ccaacgtggt catcaaggtg tgcgagttcc agttttgtaa tgatcccttc 420
ctgggcgtgt actatcacaa gaacaataag agctggatgg agtccgagtt tagagtgtat 480
tctagcgcca acaactgcac atttgagtac gtgagccagc ctttcctgat ggacctggag 540
ggcaagcagg gcaatttcaa gaacctgagg gagttcgtgt ttaagaatat cgacggctac 600
ttcaaaatct actctaagca cacccccatc aacctggtgc gcgacctgcc tcagggcttc 660
agcgccctgg agcccctggt ggatctgcct atcggcatca acatcacccg gtttcagaca 720
ctgctggccc tgcacagaag ctacctgaca cccggcgact cctctagcgg atggaccgcc 780
ggcgctgccg cctactatgt gggctacctc cagccccgga ccttcctgct gaagtacaac 840
gagaatggca ccatcacaga cgcagtggat tgcgccctgg accccctgag cgagacaaag 900
tgtacactga agtcctttac cgtggagaag ggcatctatc agacatccaa tttcagggtg 960
cagccaaccg agtctatcgt gcgctttcct aatatcacaa acctgtgccc atttggcgag 1020
gtgttcaacg caacccgctt cgccagcgtg tacgcctgga ataggaagcg gatcagcaac 1080
tgcgtggccg actatagcgt gctgtacaac tccgcctctt tcagcacctt taagtgctat 1140
ggcgtgtccc ccacaaagct gaatgacctg tgctttacca acgtctacgc cgattctttc 1200
gtgatcaggg gcgacgaggt gcgccagatc gcccccggcc agacaggcaa gatcgcagac 1260
tacaattata agctgccaga cgatttcacc ggctgcgtga tcgcctggaa cagcaacaat 1320
ctggattcca aagtgggcgg caactacaat tatctgtacc ggctgtttag aaagagcaat 1380
ctgaagccct tcgagaggga catctctaca gaaatctacc aggccggcag caccccttgc 1440
aatggcgtgg agggctttaa ctgttatttc ccactccagt cctacggctt ccagcccaca 1500
aacggcgtgg gctatcagcc ttaccgcgtg gtggtgctga gctttgagct gctgcacgcc 1560
ccagcaacag tgtgcggccc caagaagtcc accaatctgg tgaagaacaa gtgcgtgaac 1620
ttcaacttca acggcctgac cggcacaggc gtgctgaccg agtccaacaa gaagttcctg 1680
ccatttcagc agttcggcag ggacatcgca gataccacag acgccgtgcg cgacccacag 1740
accctggaga tcctggacat cacaccctgc tctttcggcg gcgtgagcgt gatcacaccc 1800
ggcaccaata caagcaacca ggtggccgtg ctgtatcagg acgtgaattg taccgaggtg 1860
cccgtggcta tccacgccga tcagctgacc ccaacatggc gggtgtacag caccggctcc 1920
aacgtcttcc agacaagagc cggatgcctg atcggagcag agcacgtgaa caattcctat 1980
gagtgcgaca tcccaatcgg cgccggcatc tgtgcctctt accagaccca gacaaactct 2040
cccagaagag cccggagcgt ggcctcccag tctatcatcg cctataccat gtccctgggc 2100
gccgagaaca gcgtggccta ctctcagaat agcatcgcca tcccaaccca gttcacaatc 2160
tctgtgacca cagagatcct gcccgtgtcc atgaccaaga catctgtgga ctgcacaatg 2220
tatatctgtg gcgattctac cgagtgcagc aacctgctgc tccagtacgg cagcttttgt 2280
acccagctga atagagccct gacaggcatc gccgtggagc aggataagaa cacacaggag 2340
gtgttcgccc aggtgaagca aatctacaag acccccccta tcaaggactt tggcggcttc 2400
caattttccc agatcctgcc tgatccatcc aagccttcta agcggagctt tatcgaggac 2460
ctgctgttca acaaggtgac cctggccgat gccggcttca tcaagcagta tggcgattgc 2520
ctgggcgaca tcgcagccag ggacctgatc tgcgcccaga agtttaatgg cctgaccgtg 2580
ctgccacccc tgctgacaga tgagatgatc gcacagtaca caagcgccct gctggccggc 2640
accatcacat ccggatggac cttcggcgca ggagccgccc tccagatccc ctttgccatg 2700
cagatggcct ataggttcaa cggcatcggc gtgacccaga atgtgctgta cgagaaccag 2760
aagctgatcg ccaatcagtt taactccgcc atcggcaaga tccaggacag cctgtcctct 2820
acagccagcg ccctgggcaa gctccaggat gtggtgaatc agaacgccca ggccctgaat 2880
accctggtga agcagctgag cagcaacttc ggcgccatct ctagcgtgct gaatgacatc 2940
ctgagccggc tggacccgcc ggaggcagag gtgcagatcg accggctgat caccggccgg 3000
ctccagagcc tccagaccta tgtgacacag cagctgatca gggccgccga gatcagggcc 3060
agcgccaatc tggcagcaac caagatgtcc gagtgcgtgc tgggccagtc taagagagtg 3120
gacttttgtg gcaagggcta tcacctgatg tccttccctc agtctgcccc acacggcgtg 3180
gtgtttctgc acgtgaccta cgtgcccgcc caggagaagc agttcaccac agcccctgcc 3240
atctgccacg atggcaaggc ccactttcca agggagggcg tgttcgtgtc ccagggcacc 3300
cactggtttg tgacacagcg caatttctac gagccccaga tcatcaccac agacaacacc 3360
ttcgtgagcg gcaactgtga cgtggtcatc ggcatcgtgc agaataccgt gtatgatcca 3420
ctccagcccg agctggacag ctttaaggag gagctggata agtatttcaa gcaacacacc 3480
tcccctgacg tggatctggg cgacatcagc ggcatccaag cctccgtggt gaacatccag 3540
aaggagatcg accgcctgaa cgaggtggct aagaatctga acgagagcct gatcgacctc 3600
caggagctgg gcaagtatga gcagtacatc aagtggccct ggtacatctg gctgggcttc 3660
atcgccggcc tgatcgccat cgtgatggtg accatcatgc tgtgctgtat gacatcctgc 3720
tgttcttgcc tgaagggctg ctgtagctgt ggctcctgct gtaagtttga cgaggatgac 3780
tctgaacctg tgctgaaggg cgtgaagctg cattacacct aa 3822
<210> 34
<211> 1273
<212> PRT
<213> Artificial Sequence
<220>
<223> S-(S2-1194) for Wuhan S-2P strain
<400> 34
Met Phe Val Phe Leu Val Leu Leu Pro Leu Val Ser Ser Gln Cys Val
1 5 10 15
Asn Leu Thr Thr Arg Thr Gln Leu Pro Pro Ala Tyr Thr Asn Ser Phe
20 25 30
Thr Arg Gly Val Tyr Tyr Pro Asp Lys Val Phe Arg Ser Ser Val Leu
35 40 45
His Ser Thr Gln Asp Leu Phe Leu Pro Phe Phe Ser Asn Val Thr Trp
50 55 60
Phe His Ala Ile His Val Ser Gly Thr Asn Gly Thr Lys Arg Phe Asp
65 70 75 80
Asn Pro Val Leu Pro Phe Asn Asp Gly Val Tyr Phe Ala Ser Thr Glu
85 90 95
Lys Ser Asn Ile Ile Arg Gly Trp Ile Phe Gly Thr Thr Leu Asp Ser
100 105 110
Lys Thr Gln Ser Leu Leu Ile Val Asn Asn Ala Thr Asn Val Val Ile
115 120 125
Lys Val Cys Glu Phe Gln Phe Cys Asn Asp Pro Phe Leu Gly Val Tyr
130 135 140
Tyr His Lys Asn Asn Lys Ser Trp Met Glu Ser Glu Phe Arg Val Tyr
145 150 155 160
Ser Ser Ala Asn Asn Cys Thr Phe Glu Tyr Val Ser Gln Pro Phe Leu
165 170 175
Met Asp Leu Glu Gly Lys Gln Gly Asn Phe Lys Asn Leu Arg Glu Phe
180 185 190
Val Phe Lys Asn Ile Asp Gly Tyr Phe Lys Ile Tyr Ser Lys His Thr
195 200 205
Pro Ile Asn Leu Val Arg Asp Leu Pro Gln Gly Phe Ser Ala Leu Glu
210 215 220
Pro Leu Val Asp Leu Pro Ile Gly Ile Asn Ile Thr Arg Phe Gln Thr
225 230 235 240
Leu Leu Ala Leu His Arg Ser Tyr Leu Thr Pro Gly Asp Ser Ser Ser
245 250 255
Gly Trp Thr Ala Gly Ala Ala Ala Tyr Tyr Val Gly Tyr Leu Gln Pro
260 265 270
Arg Thr Phe Leu Leu Lys Tyr Asn Glu Asn Gly Thr Ile Thr Asp Ala
275 280 285
Val Asp Cys Ala Leu Asp Pro Leu Ser Glu Thr Lys Cys Thr Leu Lys
290 295 300
Ser Phe Thr Val Glu Lys Gly Ile Tyr Gln Thr Ser Asn Phe Arg Val
305 310 315 320
Gln Pro Thr Glu Ser Ile Val Arg Phe Pro Asn Ile Thr Asn Leu Cys
325 330 335
Pro Phe Gly Glu Val Phe Asn Ala Thr Arg Phe Ala Ser Val Tyr Ala
340 345 350
Trp Asn Arg Lys Arg Ile Ser Asn Cys Val Ala Asp Tyr Ser Val Leu
355 360 365
Tyr Asn Ser Ala Ser Phe Ser Thr Phe Lys Cys Tyr Gly Val Ser Pro
370 375 380
Thr Lys Leu Asn Asp Leu Cys Phe Thr Asn Val Tyr Ala Asp Ser Phe
385 390 395 400
Val Ile Arg Gly Asp Glu Val Arg Gln Ile Ala Pro Gly Gln Thr Gly
405 410 415
Lys Ile Ala Asp Tyr Asn Tyr Lys Leu Pro Asp Asp Phe Thr Gly Cys
420 425 430
Val Ile Ala Trp Asn Ser Asn Asn Leu Asp Ser Lys Val Gly Gly Asn
435 440 445
Tyr Asn Tyr Leu Tyr Arg Leu Phe Arg Lys Ser Asn Leu Lys Pro Phe
450 455 460
Glu Arg Asp Ile Ser Thr Glu Ile Tyr Gln Ala Gly Ser Thr Pro Cys
465 470 475 480
Asn Gly Val Glu Gly Phe Asn Cys Tyr Phe Pro Leu Gln Ser Tyr Gly
485 490 495
Phe Gln Pro Thr Asn Gly Val Gly Tyr Gln Pro Tyr Arg Val Val Val
500 505 510
Leu Ser Phe Glu Leu Leu His Ala Pro Ala Thr Val Cys Gly Pro Lys
515 520 525
Lys Ser Thr Asn Leu Val Lys Asn Lys Cys Val Asn Phe Asn Phe Asn
530 535 540
Gly Leu Thr Gly Thr Gly Val Leu Thr Glu Ser Asn Lys Lys Phe Leu
545 550 555 560
Pro Phe Gln Gln Phe Gly Arg Asp Ile Ala Asp Thr Thr Asp Ala Val
565 570 575
Arg Asp Pro Gln Thr Leu Glu Ile Leu Asp Ile Thr Pro Cys Ser Phe
580 585 590
Gly Gly Val Ser Val Ile Thr Pro Gly Thr Asn Thr Ser Asn Gln Val
595 600 605
Ala Val Leu Tyr Gln Asp Val Asn Cys Thr Glu Val Pro Val Ala Ile
610 615 620
His Ala Asp Gln Leu Thr Pro Thr Trp Arg Val Tyr Ser Thr Gly Ser
625 630 635 640
Asn Val Phe Gln Thr Arg Ala Gly Cys Leu Ile Gly Ala Glu His Val
645 650 655
Asn Asn Ser Tyr Glu Cys Asp Ile Pro Ile Gly Ala Gly Ile Cys Ala
660 665 670
Ser Tyr Gln Thr Gln Thr Asn Ser Pro Arg Arg Ala Arg Ser Val Ala
675 680 685
Ser Gln Ser Ile Ile Ala Tyr Thr Met Ser Leu Gly Ala Glu Asn Ser
690 695 700
Val Ala Tyr Ser Gln Asn Ser Ile Ala Ile Pro Thr Gln Phe Thr Ile
705 710 715 720
Ser Val Thr Thr Glu Ile Leu Pro Val Ser Met Thr Lys Thr Ser Val
725 730 735
Asp Cys Thr Met Tyr Ile Cys Gly Asp Ser Thr Glu Cys Ser Asn Leu
740 745 750
Leu Leu Gln Tyr Gly Ser Phe Cys Thr Gln Leu Asn Arg Ala Leu Thr
755 760 765
Gly Ile Ala Val Glu Gln Asp Lys Asn Thr Gln Glu Val Phe Ala Gln
770 775 780
Val Lys Gln Ile Tyr Lys Thr Pro Pro Ile Lys Asp Phe Gly Gly Phe
785 790 795 800
Gln Phe Ser Gln Ile Leu Pro Asp Pro Ser Lys Pro Ser Lys Arg Ser
805 810 815
Phe Ile Glu Asp Leu Leu Phe Asn Lys Val Thr Leu Ala Asp Ala Gly
820 825 830
Phe Ile Lys Gln Tyr Gly Asp Cys Leu Gly Asp Ile Ala Ala Arg Asp
835 840 845
Leu Ile Cys Ala Gln Lys Phe Asn Gly Leu Thr Val Leu Pro Pro Leu
850 855 860
Leu Thr Asp Glu Met Ile Ala Gln Tyr Thr Ser Ala Leu Leu Ala Gly
865 870 875 880
Thr Ile Thr Ser Gly Trp Thr Phe Gly Ala Gly Ala Ala Leu Gln Ile
885 890 895
Pro Phe Ala Met Gln Met Ala Tyr Arg Phe Asn Gly Ile Gly Val Thr
900 905 910
Gln Asn Val Leu Tyr Glu Asn Gln Lys Leu Ile Ala Asn Gln Phe Asn
915 920 925
Ser Ala Ile Gly Lys Ile Gln Asp Ser Leu Ser Ser Thr Ala Ser Ala
930 935 940
Leu Gly Lys Leu Gln Asp Val Val Asn Gln Asn Ala Gln Ala Leu Asn
945 950 955 960
Thr Leu Val Lys Gln Leu Ser Ser Asn Phe Gly Ala Ile Ser Ser Val
965 970 975
Leu Asn Asp Ile Leu Ser Arg Leu Asp Pro Pro Glu Ala Glu Val Gln
980 985 990
Ile Asp Arg Leu Ile Thr Gly Arg Leu Gln Ser Leu Gln Thr Tyr Val
995 1000 1005
Thr Gln Gln Leu Ile Arg Ala Ala Glu Ile Arg Ala Ser Ala Asn
1010 1015 1020
Leu Ala Ala Thr Lys Met Ser Glu Cys Val Leu Gly Gln Ser Lys
1025 1030 1035
Arg Val Asp Phe Cys Gly Lys Gly Tyr His Leu Met Ser Phe Pro
1040 1045 1050
Gln Ser Ala Pro His Gly Val Val Phe Leu His Val Thr Tyr Val
1055 1060 1065
Pro Ala Gln Glu Lys Gln Phe Thr Thr Ala Pro Ala Ile Cys His
1070 1075 1080
Asp Gly Lys Ala His Phe Pro Arg Glu Gly Val Phe Val Ser Gln
1085 1090 1095
Gly Thr His Trp Phe Val Thr Gln Arg Asn Phe Tyr Glu Pro Gln
1100 1105 1110
Ile Ile Thr Thr Asp Asn Thr Phe Val Ser Gly Asn Cys Asp Val
1115 1120 1125
Val Ile Gly Ile Val Gln Asn Thr Val Tyr Asp Pro Leu Gln Pro
1130 1135 1140
Glu Leu Asp Ser Phe Lys Glu Glu Leu Asp Lys Tyr Phe Lys Gln
1145 1150 1155
His Thr Ser Pro Asp Val Asp Leu Gly Asp Ile Ser Gly Ile Gln
1160 1165 1170
Ala Ser Val Val Asn Ile Gln Lys Glu Ile Asp Arg Leu Asn Glu
1175 1180 1185
Val Ala Lys Asn Leu Asn Glu Ser Leu Ile Asp Leu Gln Glu Leu
1190 1195 1200
Gly Lys Tyr Glu Gln Tyr Ile Lys Trp Pro Trp Tyr Ile Trp Leu
1205 1210 1215
Gly Phe Ile Ala Gly Leu Ile Ala Ile Val Met Val Thr Ile Met
1220 1225 1230
Leu Cys Cys Met Thr Ser Cys Cys Ser Cys Leu Lys Gly Cys Cys
1235 1240 1245
Ser Cys Gly Ser Cys Cys Lys Phe Asp Glu Asp Asp Ser Glu Pro
1250 1255 1260
Val Leu Lys Gly Val Lys Leu His Tyr Thr
1265 1270
<210> 35
<211> 3822
<212> DNA
<213> Artificial Sequence
<220>
<223> S-(deg-RBD-801) for Wuhan S-2P strain
<400> 35
atgttcgtct tcctggtcct gctgcctctg gtctcctcac agtgcgtcaa tctgacaact 60
cggactcagc tgccacctgc ttatactaat agcttcacca gaggcgtgta ctatcctgac 120
aaggtgttta gaagctccgt gctgcactct acacaggatc tgtttctgcc attctttagc 180
aacgtgacct ggttccacgc catccacgtg agcggcacca atggcacaaa gcggttcgac 240
aatcccgtgc tgccttttaa cgatggcgtg tacttcgcct ctaccgagaa gagcaacatc 300
atcagaggct ggatctttgg caccacactg gactccaaga cacagtctct gctgatcgtg 360
aacaatgcca ccaacgtggt catcaaggtg tgcgagttcc agttttgtaa tgatcccttc 420
ctgggcgtgt actatcacaa gaacaataag agctggatgg agtccgagtt tagagtgtat 480
tctagcgcca acaactgcac atttgagtac gtgagccagc ctttcctgat ggacctggag 540
ggcaagcagg gcaatttcaa gaacctgagg gagttcgtgt ttaagaatat cgacggctac 600
ttcaaaatct actctaagca cacccccatc aacctggtgc gcgacctgcc tcagggcttc 660
agcgccctgg agcccctggt ggatctgcct atcggcatca acatcacccg gtttcagaca 720
ctgctggccc tgcacagaag ctacctgaca cccggcgact cctctagcgg atggaccgcc 780
ggcgctgccg cctactatgt gggctacctc cagccccgga ccttcctgct gaagtacaac 840
gagaatggca ccatcacaga cgcagtggat tgcgccctgg accccctgag cgagacaaag 900
tgtacactga agtcctttac cgtggagaag ggcatctatc agacatccaa tttcagggtg 960
cagccagccg aggcgatcgt gcgctttcct gaaatcacaa acctgtgccc atttggcgag 1020
gtgttcgagg caacccgctt cgccagcgtg tacgcctgga ataggaagcg gatcagcaac 1080
tgcgtggccg actatagcgt gctgtacaac tccgcctctt tcagcacctt taagtgctat 1140
ggcgtgtccc ccacaaagct gaatgacctg tgctttacca acgtctacgc cgattctttc 1200
gtgatcaggg gcgacgaggt gcgccagatc gcccccggcc agacaggcaa gatcgcagac 1260
tacaattata agctgccaga cgatttcacc ggctgcgtga tcgcctggaa cagcaacaat 1320
ctggattcca aagtgggcgg caactacaat tatctgtacc ggctgtttag aaagagcaat 1380
ctgaagccct tcgagaggga catctctaca gaaatctacc aggccggcag caccccttgc 1440
aatggcgtgg agggctttaa ctgttatttc ccactccagt cctacggctt ccagcccaca 1500
aacggcgtgg gctatcagcc ttaccgcgtg gtggtgctga gctttgagct gctgcacgcc 1560
ccagcaacag tgtgcggccc caagaagtcc accaatctgg tgaagaacaa gtgcgtgaac 1620
ttcaacttca acggcctgac cggcacaggc gtgctgaccg agtccaacaa gaagttcctg 1680
ccatttcagc agttcggcag ggacatcgca gataccacag acgccgtgcg cgacccacag 1740
accctggaga tcctggacat cacaccctgc tctttcggcg gcgtgagcgt gatcacaccc 1800
ggcaccaata caagcaacca ggtggccgtg ctgtatcagg acgtgaattg taccgaggtg 1860
cccgtggcta tccacgccga tcagctgacc ccaacatggc gggtgtacag caccggctcc 1920
aacgtcttcc agacaagagc cggatgcctg atcggagcag agcacgtgaa caattcctat 1980
gagtgcgaca tcccaatcgg cgccggcatc tgtgcctctt accagaccca gacaaactct 2040
cccagaagag cccggagcgt ggcctcccag tctatcatcg cctataccat gtccctgggc 2100
gccgagaaca gcgtggccta ctctaacaat agcatcgcca tcccaaccaa cttcacaatc 2160
tctgtgacca cagagatcct gcccgtgtcc atgaccaaga catctgtgga ctgcacaatg 2220
tatatctgtg gcgattctac cgagtgcagc aacctgctgc tccagtacgg cagcttttgt 2280
acccagctga atagagccct gacaggcatc gccgtggagc aggataagaa cacacaggag 2340
gtgttcgccc aggtgaagca aatctacaag acccccccta tcaaggactt tggcggcttc 2400
caattttccc agatcctgcc tgatccatcc aagccttcta agcggagctt tatcgaggac 2460
ctgctgttca acaaggtgac cctggccgat gccggcttca tcaagcagta tggcgattgc 2520
ctgggcgaca tcgcagccag ggacctgatc tgcgcccaga agtttaatgg cctgaccgtg 2580
ctgccacccc tgctgacaga tgagatgatc gcacagtaca caagcgccct gctggccggc 2640
accatcacat ccggatggac cttcggcgca ggagccgccc tccagatccc ctttgccatg 2700
cagatggcct ataggttcaa cggcatcggc gtgacccaga atgtgctgta cgagaaccag 2760
aagctgatcg ccaatcagtt taactccgcc atcggcaaga tccaggacag cctgtcctct 2820
acagccagcg ccctgggcaa gctccaggat gtggtgaatc agaacgccca ggccctgaat 2880
accctggtga agcagctgag cagcaacttc ggcgccatct ctagcgtgct gaatgacatc 2940
ctgagccggc tggacccgcc ggaggcagag gtgcagatcg accggctgat caccggccgg 3000
ctccagagcc tccagaccta tgtgacacag cagctgatca gggccgccga gatcagggcc 3060
agcgccaatc tggcagcaac caagatgtcc gagtgcgtgc tgggccagtc taagagagtg 3120
gacttttgtg gcaagggcta tcacctgatg tccttccctc agtctgcccc acacggcgtg 3180
gtgtttctgc acgtgaccta cgtgcccgcc caggagaaga acttcaccac agcccctgcc 3240
atctgccacg atggcaaggc ccactttcca agggagggcg tgttcgtgtc caacggcacc 3300
cactggtttg tgacacagcg caatttctac gagccccaga tcatcaccac agacaacacc 3360
ttcgtgagcg gcaactgtga cgtggtcatc ggcatcgtga acaataccgt gtatgatcca 3420
ctccagcccg agctggacag ctttaaggag gagctggata agtatttcaa gaatcacacc 3480
tcccctgacg tggatctggg cgacatcagc ggcatcaatg cctccgtggt gaacatccag 3540
aaggagatcg accgcctgaa cgaggtggct aagaatctga acgagagcct gatcgacctc 3600
caggagctgg gcaagtatga gcagtacatc aagtggccct ggtacatctg gctgggcttc 3660
atcgccggcc tgatcgccat cgtgatggtg accatcatgc tgtgctgtat gacatcctgc 3720
tgttcttgcc tgaagggctg ctgtagctgt ggctcctgct gtaagtttga cgaggatgac 3780
tctgaacctg tgctgaaggg cgtgaagctg cattacacct aa 3822
<210> 36
<211> 1273
<212> PRT
<213> Artificial Sequence
<220>
<223> S-(deg-RBD-801) for Wuhan S-2P strain
<400> 36
Met Phe Val Phe Leu Val Leu Leu Pro Leu Val Ser Ser Gln Cys Val
1 5 10 15
Asn Leu Thr Thr Arg Thr Gln Leu Pro Pro Ala Tyr Thr Asn Ser Phe
20 25 30
Thr Arg Gly Val Tyr Tyr Pro Asp Lys Val Phe Arg Ser Ser Val Leu
35 40 45
His Ser Thr Gln Asp Leu Phe Leu Pro Phe Phe Ser Asn Val Thr Trp
50 55 60
Phe His Ala Ile His Val Ser Gly Thr Asn Gly Thr Lys Arg Phe Asp
65 70 75 80
Asn Pro Val Leu Pro Phe Asn Asp Gly Val Tyr Phe Ala Ser Thr Glu
85 90 95
Lys Ser Asn Ile Ile Arg Gly Trp Ile Phe Gly Thr Thr Leu Asp Ser
100 105 110
Lys Thr Gln Ser Leu Leu Ile Val Asn Asn Ala Thr Asn Val Val Ile
115 120 125
Lys Val Cys Glu Phe Gln Phe Cys Asn Asp Pro Phe Leu Gly Val Tyr
130 135 140
Tyr His Lys Asn Asn Lys Ser Trp Met Glu Ser Glu Phe Arg Val Tyr
145 150 155 160
Ser Ser Ala Asn Asn Cys Thr Phe Glu Tyr Val Ser Gln Pro Phe Leu
165 170 175
Met Asp Leu Glu Gly Lys Gln Gly Asn Phe Lys Asn Leu Arg Glu Phe
180 185 190
Val Phe Lys Asn Ile Asp Gly Tyr Phe Lys Ile Tyr Ser Lys His Thr
195 200 205
Pro Ile Asn Leu Val Arg Asp Leu Pro Gln Gly Phe Ser Ala Leu Glu
210 215 220
Pro Leu Val Asp Leu Pro Ile Gly Ile Asn Ile Thr Arg Phe Gln Thr
225 230 235 240
Leu Leu Ala Leu His Arg Ser Tyr Leu Thr Pro Gly Asp Ser Ser Ser
245 250 255
Gly Trp Thr Ala Gly Ala Ala Ala Tyr Tyr Val Gly Tyr Leu Gln Pro
260 265 270
Arg Thr Phe Leu Leu Lys Tyr Asn Glu Asn Gly Thr Ile Thr Asp Ala
275 280 285
Val Asp Cys Ala Leu Asp Pro Leu Ser Glu Thr Lys Cys Thr Leu Lys
290 295 300
Ser Phe Thr Val Glu Lys Gly Ile Tyr Gln Thr Ser Asn Phe Arg Val
305 310 315 320
Gln Pro Ala Glu Ala Ile Val Arg Phe Pro Gln Ile Thr Asn Leu Cys
325 330 335
Pro Phe Gly Glu Val Phe Gln Ala Thr Arg Phe Ala Ser Val Tyr Ala
340 345 350
Trp Asn Arg Lys Arg Ile Ser Asn Cys Val Ala Asp Tyr Ser Val Leu
355 360 365
Tyr Asn Ser Ala Ser Phe Ser Thr Phe Lys Cys Tyr Gly Val Ser Pro
370 375 380
Thr Lys Leu Asn Asp Leu Cys Phe Thr Asn Val Tyr Ala Asp Ser Phe
385 390 395 400
Val Ile Arg Gly Asp Glu Val Arg Gln Ile Ala Pro Gly Gln Thr Gly
405 410 415
Lys Ile Ala Asp Tyr Asn Tyr Lys Leu Pro Asp Asp Phe Thr Gly Cys
420 425 430
Val Ile Ala Trp Asn Ser Asn Asn Leu Asp Ser Lys Val Gly Gly Asn
435 440 445
Tyr Asn Tyr Leu Tyr Arg Leu Phe Arg Lys Ser Asn Leu Lys Pro Phe
450 455 460
Glu Arg Asp Ile Ser Thr Glu Ile Tyr Gln Ala Gly Ser Thr Pro Cys
465 470 475 480
Asn Gly Val Glu Gly Phe Asn Cys Tyr Phe Pro Leu Gln Ser Tyr Gly
485 490 495
Phe Gln Pro Thr Asn Gly Val Gly Tyr Gln Pro Tyr Arg Val Val Val
500 505 510
Leu Ser Phe Glu Leu Leu His Ala Pro Ala Thr Val Cys Gly Pro Lys
515 520 525
Lys Ser Thr Asn Leu Val Lys Asn Lys Cys Val Asn Phe Asn Phe Asn
530 535 540
Gly Leu Thr Gly Thr Gly Val Leu Thr Glu Ser Asn Lys Lys Phe Leu
545 550 555 560
Pro Phe Gln Gln Phe Gly Arg Asp Ile Ala Asp Thr Thr Asp Ala Val
565 570 575
Arg Asp Pro Gln Thr Leu Glu Ile Leu Asp Ile Thr Pro Cys Ser Phe
580 585 590
Gly Gly Val Ser Val Ile Thr Pro Gly Thr Asn Thr Ser Asn Gln Val
595 600 605
Ala Val Leu Tyr Gln Asp Val Asn Cys Thr Glu Val Pro Val Ala Ile
610 615 620
His Ala Asp Gln Leu Thr Pro Thr Trp Arg Val Tyr Ser Thr Gly Ser
625 630 635 640
Asn Val Phe Gln Thr Arg Ala Gly Cys Leu Ile Gly Ala Glu His Val
645 650 655
Asn Asn Ser Tyr Glu Cys Asp Ile Pro Ile Gly Ala Gly Ile Cys Ala
660 665 670
Ser Tyr Gln Thr Gln Thr Asn Ser Pro Arg Arg Ala Arg Ser Val Ala
675 680 685
Ser Gln Ser Ile Ile Ala Tyr Thr Met Ser Leu Gly Ala Glu Asn Ser
690 695 700
Val Ala Tyr Ser Asn Asn Ser Ile Ala Ile Pro Thr Asn Phe Thr Ile
705 710 715 720
Ser Val Thr Thr Glu Ile Leu Pro Val Ser Met Thr Lys Thr Ser Val
725 730 735
Asp Cys Thr Met Tyr Ile Cys Gly Asp Ser Thr Glu Cys Ser Asn Leu
740 745 750
Leu Leu Gln Tyr Gly Ser Phe Cys Thr Gln Leu Asn Arg Ala Leu Thr
755 760 765
Gly Ile Ala Val Glu Gln Asp Lys Asn Thr Gln Glu Val Phe Ala Gln
770 775 780
Val Lys Gln Ile Tyr Lys Thr Pro Pro Ile Lys Asp Phe Gly Gly Phe
785 790 795 800
Gln Phe Ser Gln Ile Leu Pro Asp Pro Ser Lys Pro Ser Lys Arg Ser
805 810 815
Phe Ile Glu Asp Leu Leu Phe Asn Lys Val Thr Leu Ala Asp Ala Gly
820 825 830
Phe Ile Lys Gln Tyr Gly Asp Cys Leu Gly Asp Ile Ala Ala Arg Asp
835 840 845
Leu Ile Cys Ala Gln Lys Phe Asn Gly Leu Thr Val Leu Pro Pro Leu
850 855 860
Leu Thr Asp Glu Met Ile Ala Gln Tyr Thr Ser Ala Leu Leu Ala Gly
865 870 875 880
Thr Ile Thr Ser Gly Trp Thr Phe Gly Ala Gly Ala Ala Leu Gln Ile
885 890 895
Pro Phe Ala Met Gln Met Ala Tyr Arg Phe Asn Gly Ile Gly Val Thr
900 905 910
Gln Asn Val Leu Tyr Glu Asn Gln Lys Leu Ile Ala Asn Gln Phe Asn
915 920 925
Ser Ala Ile Gly Lys Ile Gln Asp Ser Leu Ser Ser Thr Ala Ser Ala
930 935 940
Leu Gly Lys Leu Gln Asp Val Val Asn Gln Asn Ala Gln Ala Leu Asn
945 950 955 960
Thr Leu Val Lys Gln Leu Ser Ser Asn Phe Gly Ala Ile Ser Ser Val
965 970 975
Leu Asn Asp Ile Leu Ser Arg Leu Asp Pro Pro Glu Ala Glu Val Gln
980 985 990
Ile Asp Arg Leu Ile Thr Gly Arg Leu Gln Ser Leu Gln Thr Tyr Val
995 1000 1005
Thr Gln Gln Leu Ile Arg Ala Ala Glu Ile Arg Ala Ser Ala Asn
1010 1015 1020
Leu Ala Ala Thr Lys Met Ser Glu Cys Val Leu Gly Gln Ser Lys
1025 1030 1035
Arg Val Asp Phe Cys Gly Lys Gly Tyr His Leu Met Ser Phe Pro
1040 1045 1050
Gln Ser Ala Pro His Gly Val Val Phe Leu His Val Thr Tyr Val
1055 1060 1065
Pro Ala Gln Glu Lys Asn Phe Thr Thr Ala Pro Ala Ile Cys His
1070 1075 1080
Asp Gly Lys Ala His Phe Pro Arg Glu Gly Val Phe Val Ser Asn
1085 1090 1095
Gly Thr His Trp Phe Val Thr Gln Arg Asn Phe Tyr Glu Pro Gln
1100 1105 1110
Ile Ile Thr Thr Asp Asn Thr Phe Val Ser Gly Asn Cys Asp Val
1115 1120 1125
Val Ile Gly Ile Val Asn Asn Thr Val Tyr Asp Pro Leu Gln Pro
1130 1135 1140
Glu Leu Asp Ser Phe Lys Glu Glu Leu Asp Lys Tyr Phe Lys Asn
1145 1150 1155
His Thr Ser Pro Asp Val Asp Leu Gly Asp Ile Ser Gly Ile Asn
1160 1165 1170
Ala Ser Val Val Asn Ile Gln Lys Glu Ile Asp Arg Leu Asn Glu
1175 1180 1185
Val Ala Lys Asn Leu Asn Glu Ser Leu Ile Asp Leu Gln Glu Leu
1190 1195 1200
Gly Lys Tyr Glu Gln Tyr Ile Lys Trp Pro Trp Tyr Ile Trp Leu
1205 1210 1215
Gly Phe Ile Ala Gly Leu Ile Ala Ile Val Met Val Thr Ile Met
1220 1225 1230
Leu Cys Cys Met Thr Ser Cys Cys Ser Cys Leu Lys Gly Cys Cys
1235 1240 1245
Ser Cys Gly Ser Cys Cys Lys Phe Asp Glu Asp Asp Ser Glu Pro
1250 1255 1260
Val Leu Lys Gly Val Lys Leu His Tyr Thr
1265 1270
<210> 37
<211> 3822
<212> DNA
<213> Artificial Sequence
<220>
<223> S-(deg-RBD-1194) for Wuhan S-2P strain
<400> 37
atgttcgtct tcctggtcct gctgcctctg gtctcctcac agtgcgtcaa tctgacaact 60
cggactcagc tgccacctgc ttatactaat agcttcacca gaggcgtgta ctatcctgac 120
aaggtgttta gaagctccgt gctgcactct acacaggatc tgtttctgcc attctttagc 180
aacgtgacct ggttccacgc catccacgtg agcggcacca atggcacaaa gcggttcgac 240
aatcccgtgc tgccttttaa cgatggcgtg tacttcgcct ctaccgagaa gagcaacatc 300
atcagaggct ggatctttgg caccacactg gactccaaga cacagtctct gctgatcgtg 360
aacaatgcca ccaacgtggt catcaaggtg tgcgagttcc agttttgtaa tgatcccttc 420
ctgggcgtgt actatcacaa gaacaataag agctggatgg agtccgagtt tagagtgtat 480
tctagcgcca acaactgcac atttgagtac gtgagccagc ctttcctgat ggacctggag 540
ggcaagcagg gcaatttcaa gaacctgagg gagttcgtgt ttaagaatat cgacggctac 600
ttcaaaatct actctaagca cacccccatc aacctggtgc gcgacctgcc tcagggcttc 660
agcgccctgg agcccctggt ggatctgcct atcggcatca acatcacccg gtttcagaca 720
ctgctggccc tgcacagaag ctacctgaca cccggcgact cctctagcgg atggaccgcc 780
ggcgctgccg cctactatgt gggctacctc cagccccgga ccttcctgct gaagtacaac 840
gagaatggca ccatcacaga cgcagtggat tgcgccctgg accccctgag cgagacaaag 900
tgtacactga agtcctttac cgtggagaag ggcatctatc agacatccaa tttcagggtg 960
cagccagccg aggcgatcgt gcgctttcct gaaatcacaa acctgtgccc atttggcgag 1020
gtgttcgagg caacccgctt cgccagcgtg tacgcctgga ataggaagcg gatcagcaac 1080
tgcgtggccg actatagcgt gctgtacaac tccgcctctt tcagcacctt taagtgctat 1140
ggcgtgtccc ccacaaagct gaatgacctg tgctttacca acgtctacgc cgattctttc 1200
gtgatcaggg gcgacgaggt gcgccagatc gcccccggcc agacaggcaa gatcgcagac 1260
tacaattata agctgccaga cgatttcacc ggctgcgtga tcgcctggaa cagcaacaat 1320
ctggattcca aagtgggcgg caactacaat tatctgtacc ggctgtttag aaagagcaat 1380
ctgaagccct tcgagaggga catctctaca gaaatctacc aggccggcag caccccttgc 1440
aatggcgtgg agggctttaa ctgttatttc ccactccagt cctacggctt ccagcccaca 1500
aacggcgtgg gctatcagcc ttaccgcgtg gtggtgctga gctttgagct gctgcacgcc 1560
ccagcaacag tgtgcggccc caagaagtcc accaatctgg tgaagaacaa gtgcgtgaac 1620
ttcaacttca acggcctgac cggcacaggc gtgctgaccg agtccaacaa gaagttcctg 1680
ccatttcagc agttcggcag ggacatcgca gataccacag acgccgtgcg cgacccacag 1740
accctggaga tcctggacat cacaccctgc tctttcggcg gcgtgagcgt gatcacaccc 1800
ggcaccaata caagcaacca ggtggccgtg ctgtatcagg acgtgaattg taccgaggtg 1860
cccgtggcta tccacgccga tcagctgacc ccaacatggc gggtgtacag caccggctcc 1920
aacgtcttcc agacaagagc cggatgcctg atcggagcag agcacgtgaa caattcctat 1980
gagtgcgaca tcccaatcgg cgccggcatc tgtgcctctt accagaccca gacaaactct 2040
cccagaagag cccggagcgt ggcctcccag tctatcatcg cctataccat gtccctgggc 2100
gccgagaaca gcgtggccta ctctaacaat agcatcgcca tcccaaccaa cttcacaatc 2160
tctgtgacca cagagatcct gcccgtgtcc atgaccaaga catctgtgga ctgcacaatg 2220
tatatctgtg gcgattctac cgagtgcagc aacctgctgc tccagtacgg cagcttttgt 2280
acccagctga atagagccct gacaggcatc gccgtggagc aggataagaa cacacaggag 2340
gtgttcgccc aggtgaagca aatctacaag acccccccta tcaaggactt tggcggcttc 2400
aatttttccc agatcctgcc tgatccatcc aagccttcta agcggagctt tatcgaggac 2460
ctgctgttca acaaggtgac cctggccgat gccggcttca tcaagcagta tggcgattgc 2520
ctgggcgaca tcgcagccag ggacctgatc tgcgcccaga agtttaatgg cctgaccgtg 2580
ctgccacccc tgctgacaga tgagatgatc gcacagtaca caagcgccct gctggccggc 2640
accatcacat ccggatggac cttcggcgca ggagccgccc tccagatccc ctttgccatg 2700
cagatggcct ataggttcaa cggcatcggc gtgacccaga atgtgctgta cgagaaccag 2760
aagctgatcg ccaatcagtt taactccgcc atcggcaaga tccaggacag cctgtcctct 2820
acagccagcg ccctgggcaa gctccaggat gtggtgaatc agaacgccca ggccctgaat 2880
accctggtga agcagctgag cagcaacttc ggcgccatct ctagcgtgct gaatgacatc 2940
ctgagccggc tggacccgcc ggaggcagag gtgcagatcg accggctgat caccggccgg 3000
ctccagagcc tccagaccta tgtgacacag cagctgatca gggccgccga gatcagggcc 3060
agcgccaatc tggcagcaac caagatgtcc gagtgcgtgc tgggccagtc taagagagtg 3120
gacttttgtg gcaagggcta tcacctgatg tccttccctc agtctgcccc acacggcgtg 3180
gtgtttctgc acgtgaccta cgtgcccgcc caggagaaga acttcaccac agcccctgcc 3240
atctgccacg atggcaaggc ccactttcca agggagggcg tgttcgtgtc caacggcacc 3300
cactggtttg tgacacagcg caatttctac gagccccaga tcatcaccac agacaacacc 3360
ttcgtgagcg gcaactgtga cgtggtcatc ggcatcgtga acaataccgt gtatgatcca 3420
ctccagcccg agctggacag ctttaaggag gagctggata agtatttcaa gaatcacacc 3480
tcccctgacg tggatctggg cgacatcagc ggcatcaatg cctccgtggt gaacatccag 3540
aaggagatcg accgcctgaa cgaggtggct aagaatctgc aggagagcct gatcgacctc 3600
caggagctgg gcaagtatga gcagtacatc aagtggccct ggtacatctg gctgggcttc 3660
atcgccggcc tgatcgccat cgtgatggtg accatcatgc tgtgctgtat gacatcctgc 3720
tgttcttgcc tgaagggctg ctgtagctgt ggctcctgct gtaagtttga cgaggatgac 3780
tctgaacctg tgctgaaggg cgtgaagctg cattacacct aa 3822
<210> 38
<211> 1273
<212> PRT
<213> Artificial Sequence
<220>
<223> S-(deg-RBD-1194) for Wuhan S-2P strain
<400> 38
Met Phe Val Phe Leu Val Leu Leu Pro Leu Val Ser Ser Gln Cys Val
1 5 10 15
Asn Leu Thr Thr Arg Thr Gln Leu Pro Pro Ala Tyr Thr Asn Ser Phe
20 25 30
Thr Arg Gly Val Tyr Tyr Pro Asp Lys Val Phe Arg Ser Ser Val Leu
35 40 45
His Ser Thr Gln Asp Leu Phe Leu Pro Phe Phe Ser Asn Val Thr Trp
50 55 60
Phe His Ala Ile His Val Ser Gly Thr Asn Gly Thr Lys Arg Phe Asp
65 70 75 80
Asn Pro Val Leu Pro Phe Asn Asp Gly Val Tyr Phe Ala Ser Thr Glu
85 90 95
Lys Ser Asn Ile Ile Arg Gly Trp Ile Phe Gly Thr Thr Leu Asp Ser
100 105 110
Lys Thr Gln Ser Leu Leu Ile Val Asn Asn Ala Thr Asn Val Val Ile
115 120 125
Lys Val Cys Glu Phe Gln Phe Cys Asn Asp Pro Phe Leu Gly Val Tyr
130 135 140
Tyr His Lys Asn Asn Lys Ser Trp Met Glu Ser Glu Phe Arg Val Tyr
145 150 155 160
Ser Ser Ala Asn Asn Cys Thr Phe Glu Tyr Val Ser Gln Pro Phe Leu
165 170 175
Met Asp Leu Glu Gly Lys Gln Gly Asn Phe Lys Asn Leu Arg Glu Phe
180 185 190
Val Phe Lys Asn Ile Asp Gly Tyr Phe Lys Ile Tyr Ser Lys His Thr
195 200 205
Pro Ile Asn Leu Val Arg Asp Leu Pro Gln Gly Phe Ser Ala Leu Glu
210 215 220
Pro Leu Val Asp Leu Pro Ile Gly Ile Asn Ile Thr Arg Phe Gln Thr
225 230 235 240
Leu Leu Ala Leu His Arg Ser Tyr Leu Thr Pro Gly Asp Ser Ser Ser
245 250 255
Gly Trp Thr Ala Gly Ala Ala Ala Tyr Tyr Val Gly Tyr Leu Gln Pro
260 265 270
Arg Thr Phe Leu Leu Lys Tyr Asn Glu Asn Gly Thr Ile Thr Asp Ala
275 280 285
Val Asp Cys Ala Leu Asp Pro Leu Ser Glu Thr Lys Cys Thr Leu Lys
290 295 300
Ser Phe Thr Val Glu Lys Gly Ile Tyr Gln Thr Ser Asn Phe Arg Val
305 310 315 320
Gln Pro Ala Glu Ala Ile Val Arg Phe Pro Gln Ile Thr Asn Leu Cys
325 330 335
Pro Phe Gly Glu Val Phe Gln Ala Thr Arg Phe Ala Ser Val Tyr Ala
340 345 350
Trp Asn Arg Lys Arg Ile Ser Asn Cys Val Ala Asp Tyr Ser Val Leu
355 360 365
Tyr Asn Ser Ala Ser Phe Ser Thr Phe Lys Cys Tyr Gly Val Ser Pro
370 375 380
Thr Lys Leu Asn Asp Leu Cys Phe Thr Asn Val Tyr Ala Asp Ser Phe
385 390 395 400
Val Ile Arg Gly Asp Glu Val Arg Gln Ile Ala Pro Gly Gln Thr Gly
405 410 415
Lys Ile Ala Asp Tyr Asn Tyr Lys Leu Pro Asp Asp Phe Thr Gly Cys
420 425 430
Val Ile Ala Trp Asn Ser Asn Asn Leu Asp Ser Lys Val Gly Gly Asn
435 440 445
Tyr Asn Tyr Leu Tyr Arg Leu Phe Arg Lys Ser Asn Leu Lys Pro Phe
450 455 460
Glu Arg Asp Ile Ser Thr Glu Ile Tyr Gln Ala Gly Ser Thr Pro Cys
465 470 475 480
Asn Gly Val Glu Gly Phe Asn Cys Tyr Phe Pro Leu Gln Ser Tyr Gly
485 490 495
Phe Gln Pro Thr Asn Gly Val Gly Tyr Gln Pro Tyr Arg Val Val Val
500 505 510
Leu Ser Phe Glu Leu Leu His Ala Pro Ala Thr Val Cys Gly Pro Lys
515 520 525
Lys Ser Thr Asn Leu Val Lys Asn Lys Cys Val Asn Phe Asn Phe Asn
530 535 540
Gly Leu Thr Gly Thr Gly Val Leu Thr Glu Ser Asn Lys Lys Phe Leu
545 550 555 560
Pro Phe Gln Gln Phe Gly Arg Asp Ile Ala Asp Thr Thr Asp Ala Val
565 570 575
Arg Asp Pro Gln Thr Leu Glu Ile Leu Asp Ile Thr Pro Cys Ser Phe
580 585 590
Gly Gly Val Ser Val Ile Thr Pro Gly Thr Asn Thr Ser Asn Gln Val
595 600 605
Ala Val Leu Tyr Gln Asp Val Asn Cys Thr Glu Val Pro Val Ala Ile
610 615 620
His Ala Asp Gln Leu Thr Pro Thr Trp Arg Val Tyr Ser Thr Gly Ser
625 630 635 640
Asn Val Phe Gln Thr Arg Ala Gly Cys Leu Ile Gly Ala Glu His Val
645 650 655
Asn Asn Ser Tyr Glu Cys Asp Ile Pro Ile Gly Ala Gly Ile Cys Ala
660 665 670
Ser Tyr Gln Thr Gln Thr Asn Ser Pro Arg Arg Ala Arg Ser Val Ala
675 680 685
Ser Gln Ser Ile Ile Ala Tyr Thr Met Ser Leu Gly Ala Glu Asn Ser
690 695 700
Val Ala Tyr Ser Asn Asn Ser Ile Ala Ile Pro Thr Asn Phe Thr Ile
705 710 715 720
Ser Val Thr Thr Glu Ile Leu Pro Val Ser Met Thr Lys Thr Ser Val
725 730 735
Asp Cys Thr Met Tyr Ile Cys Gly Asp Ser Thr Glu Cys Ser Asn Leu
740 745 750
Leu Leu Gln Tyr Gly Ser Phe Cys Thr Gln Leu Asn Arg Ala Leu Thr
755 760 765
Gly Ile Ala Val Glu Gln Asp Lys Asn Thr Gln Glu Val Phe Ala Gln
770 775 780
Val Lys Gln Ile Tyr Lys Thr Pro Pro Ile Lys Asp Phe Gly Gly Phe
785 790 795 800
Asn Phe Ser Gln Ile Leu Pro Asp Pro Ser Lys Pro Ser Lys Arg Ser
805 810 815
Phe Ile Glu Asp Leu Leu Phe Asn Lys Val Thr Leu Ala Asp Ala Gly
820 825 830
Phe Ile Lys Gln Tyr Gly Asp Cys Leu Gly Asp Ile Ala Ala Arg Asp
835 840 845
Leu Ile Cys Ala Gln Lys Phe Asn Gly Leu Thr Val Leu Pro Pro Leu
850 855 860
Leu Thr Asp Glu Met Ile Ala Gln Tyr Thr Ser Ala Leu Leu Ala Gly
865 870 875 880
Thr Ile Thr Ser Gly Trp Thr Phe Gly Ala Gly Ala Ala Leu Gln Ile
885 890 895
Pro Phe Ala Met Gln Met Ala Tyr Arg Phe Asn Gly Ile Gly Val Thr
900 905 910
Gln Asn Val Leu Tyr Glu Asn Gln Lys Leu Ile Ala Asn Gln Phe Asn
915 920 925
Ser Ala Ile Gly Lys Ile Gln Asp Ser Leu Ser Ser Thr Ala Ser Ala
930 935 940
Leu Gly Lys Leu Gln Asp Val Val Asn Gln Asn Ala Gln Ala Leu Asn
945 950 955 960
Thr Leu Val Lys Gln Leu Ser Ser Asn Phe Gly Ala Ile Ser Ser Val
965 970 975
Leu Asn Asp Ile Leu Ser Arg Leu Asp Pro Pro Glu Ala Glu Val Gln
980 985 990
Ile Asp Arg Leu Ile Thr Gly Arg Leu Gln Ser Leu Gln Thr Tyr Val
995 1000 1005
Thr Gln Gln Leu Ile Arg Ala Ala Glu Ile Arg Ala Ser Ala Asn
1010 1015 1020
Leu Ala Ala Thr Lys Met Ser Glu Cys Val Leu Gly Gln Ser Lys
1025 1030 1035
Arg Val Asp Phe Cys Gly Lys Gly Tyr His Leu Met Ser Phe Pro
1040 1045 1050
Gln Ser Ala Pro His Gly Val Val Phe Leu His Val Thr Tyr Val
1055 1060 1065
Pro Ala Gln Glu Lys Asn Phe Thr Thr Ala Pro Ala Ile Cys His
1070 1075 1080
Asp Gly Lys Ala His Phe Pro Arg Glu Gly Val Phe Val Ser Asn
1085 1090 1095
Gly Thr His Trp Phe Val Thr Gln Arg Asn Phe Tyr Glu Pro Gln
1100 1105 1110
Ile Ile Thr Thr Asp Asn Thr Phe Val Ser Gly Asn Cys Asp Val
1115 1120 1125
Val Ile Gly Ile Val Asn Asn Thr Val Tyr Asp Pro Leu Gln Pro
1130 1135 1140
Glu Leu Asp Ser Phe Lys Glu Glu Leu Asp Lys Tyr Phe Lys Asn
1145 1150 1155
His Thr Ser Pro Asp Val Asp Leu Gly Asp Ile Ser Gly Ile Asn
1160 1165 1170
Ala Ser Val Val Asn Ile Gln Lys Glu Ile Asp Arg Leu Asn Glu
1175 1180 1185
Val Ala Lys Asn Leu Gln Glu Ser Leu Ile Asp Leu Gln Glu Leu
1190 1195 1200
Gly Lys Tyr Glu Gln Tyr Ile Lys Trp Pro Trp Tyr Ile Trp Leu
1205 1210 1215
Gly Phe Ile Ala Gly Leu Ile Ala Ile Val Met Val Thr Ile Met
1220 1225 1230
Leu Cys Cys Met Thr Ser Cys Cys Ser Cys Leu Lys Gly Cys Cys
1235 1240 1245
Ser Cys Gly Ser Cys Cys Lys Phe Asp Glu Asp Asp Ser Glu Pro
1250 1255 1260
Val Leu Lys Gly Val Lys Leu His Tyr Thr
1265 1270
<210> 39
<211> 3822
<212> DNA
<213> Artificial Sequence
<220>
<223> S-(deg-RBD-122-165-234) for Wuhan S-2P strain
<400> 39
atgttcgtct tcctggtcct gctgcctctg gtctcctcac agtgcgtcaa tctgacaact 60
cggactcagc tgccacctgc ttatactaat agcttcacca gaggcgtgta ctatcctgac 120
aaggtgttta gaagctccgt gctgcactct acacaggatc tgtttctgcc attctttagc 180
aacgtgacct ggttccacgc catccacgtg agcggcacca atggcacaaa gcggttcgac 240
aatcccgtgc tgccttttaa cgatggcgtg tacttcgcct ctaccgagaa gagcaacatc 300
atcagaggct ggatctttgg caccacactg gactccaaga cacagtctct gctgatcgtg 360
aaccaagcca ccaacgtggt catcaaggtg tgcgagttcc agttttgtaa tgatcccttc 420
ctgggcgtgt actatcacaa gaacaataag agctggatgg agtccgagtt tagagtgtat 480
tctagcgcca accagtgcac atttgagtac gtgagccagc ctttcctgat ggacctggag 540
ggcaagcagg gcaatttcaa gaacctgagg gagttcgtgt ttaagaatat cgacggctac 600
ttcaaaatct actctaagca cacccccatc aacctggtgc gcgacctgcc tcagggcttc 660
agcgccctgg agcccctggt ggatctgcct atcggcatcc agatcacccg gtttcagaca 720
ctgctggccc tgcacagaag ctacctgaca cccggcgact cctctagcgg atggaccgcc 780
ggcgctgccg cctactatgt gggctacctc cagccccgga ccttcctgct gaagtacaac 840
gagaatggca ccatcacaga cgcagtggat tgcgccctgg accccctgag cgagacaaag 900
tgtacactga agtcctttac cgtggagaag ggcatctatc agacatccaa tttcagggtg 960
cagccagccg aggcgatcgt gcgctttcct gaaatcacaa acctgtgccc atttggcgag 1020
gtgttcgagg caacccgctt cgccagcgtg tacgcctgga ataggaagcg gatcagcaac 1080
tgcgtggccg actatagcgt gctgtacaac tccgcctctt tcagcacctt taagtgctat 1140
ggcgtgtccc ccacaaagct gaatgacctg tgctttacca acgtctacgc cgattctttc 1200
gtgatcaggg gcgacgaggt gcgccagatc gcccccggcc agacaggcaa gatcgcagac 1260
tacaattata agctgccaga cgatttcacc ggctgcgtga tcgcctggaa cagcaacaat 1320
ctggattcca aagtgggcgg caactacaat tatctgtacc ggctgtttag aaagagcaat 1380
ctgaagccct tcgagaggga catctctaca gaaatctacc aggccggcag caccccttgc 1440
aatggcgtgg agggctttaa ctgttatttc ccactccagt cctacggctt ccagcccaca 1500
aacggcgtgg gctatcagcc ttaccgcgtg gtggtgctga gctttgagct gctgcacgcc 1560
ccagcaacag tgtgcggccc caagaagtcc accaatctgg tgaagaacaa gtgcgtgaac 1620
ttcaacttca acggcctgac cggcacaggc gtgctgaccg agtccaacaa gaagttcctg 1680
ccatttcagc agttcggcag ggacatcgca gataccacag acgccgtgcg cgacccacag 1740
accctggaga tcctggacat cacaccctgc tctttcggcg gcgtgagcgt gatcacaccc 1800
ggcaccaata caagcaacca ggtggccgtg ctgtatcagg acgtgaattg taccgaggtg 1860
cccgtggcta tccacgccga tcagctgacc ccaacatggc gggtgtacag caccggctcc 1920
aacgtcttcc agacaagagc cggatgcctg atcggagcag agcacgtgaa caattcctat 1980
gagtgcgaca tcccaatcgg cgccggcatc tgtgcctctt accagaccca gacaaactct 2040
cccagaagag cccggagcgt ggcctcccag tctatcatcg cctataccat gtccctgggc 2100
gccgagaaca gcgtggccta ctctaacaat agcatcgcca tcccaaccaa cttcacaatc 2160
tctgtgacca cagagatcct gcccgtgtcc atgaccaaga catctgtgga ctgcacaatg 2220
tatatctgtg gcgattctac cgagtgcagc aacctgctgc tccagtacgg cagcttttgt 2280
acccagctga atagagccct gacaggcatc gccgtggagc aggataagaa cacacaggag 2340
gtgttcgccc aggtgaagca aatctacaag acccccccta tcaaggactt tggcggcttc 2400
aatttttccc agatcctgcc tgatccatcc aagccttcta agcggagctt tatcgaggac 2460
ctgctgttca acaaggtgac cctggccgat gccggcttca tcaagcagta tggcgattgc 2520
ctgggcgaca tcgcagccag ggacctgatc tgcgcccaga agtttaatgg cctgaccgtg 2580
ctgccacccc tgctgacaga tgagatgatc gcacagtaca caagcgccct gctggccggc 2640
accatcacat ccggatggac cttcggcgca ggagccgccc tccagatccc ctttgccatg 2700
cagatggcct ataggttcaa cggcatcggc gtgacccaga atgtgctgta cgagaaccag 2760
aagctgatcg ccaatcagtt taactccgcc atcggcaaga tccaggacag cctgtcctct 2820
acagccagcg ccctgggcaa gctccaggat gtggtgaatc agaacgccca ggccctgaat 2880
accctggtga agcagctgag cagcaacttc ggcgccatct ctagcgtgct gaatgacatc 2940
ctgagccggc tggacccgcc ggaggcagag gtgcagatcg accggctgat caccggccgg 3000
ctccagagcc tccagaccta tgtgacacag cagctgatca gggccgccga gatcagggcc 3060
agcgccaatc tggcagcaac caagatgtcc gagtgcgtgc tgggccagtc taagagagtg 3120
gacttttgtg gcaagggcta tcacctgatg tccttccctc agtctgcccc acacggcgtg 3180
gtgtttctgc acgtgaccta cgtgcccgcc caggagaaga acttcaccac agcccctgcc 3240
atctgccacg atggcaaggc ccactttcca agggagggcg tgttcgtgtc caacggcacc 3300
cactggtttg tgacacagcg caatttctac gagccccaga tcatcaccac agacaacacc 3360
ttcgtgagcg gcaactgtga cgtggtcatc ggcatcgtga acaataccgt gtatgatcca 3420
ctccagcccg agctggacag ctttaaggag gagctggata agtatttcaa gaatcacacc 3480
tcccctgacg tggatctggg cgacatcagc ggcatcaatg cctccgtggt gaacatccag 3540
aaggagatcg accgcctgaa cgaggtggct aagaatctga acgagagcct gatcgacctc 3600
caggagctgg gcaagtatga gcagtacatc aagtggccct ggtacatctg gctgggcttc 3660
atcgccggcc tgatcgccat cgtgatggtg accatcatgc tgtgctgtat gacatcctgc 3720
tgttcttgcc tgaagggctg ctgtagctgt ggctcctgct gtaagtttga cgaggatgac 3780
tctgaacctg tgctgaaggg cgtgaagctg cattacacct aa 3822
<210> 40
<211> 1273
<212> PRT
<213> Artificial Sequence
<220>
<223> S-(deg-RBD-122-165-234) for Wuhan S-2P strain
<400> 40
Met Phe Val Phe Leu Val Leu Leu Pro Leu Val Ser Ser Gln Cys Val
1 5 10 15
Asn Leu Thr Thr Arg Thr Gln Leu Pro Pro Ala Tyr Thr Asn Ser Phe
20 25 30
Thr Arg Gly Val Tyr Tyr Pro Asp Lys Val Phe Arg Ser Ser Val Leu
35 40 45
His Ser Thr Gln Asp Leu Phe Leu Pro Phe Phe Ser Asn Val Thr Trp
50 55 60
Phe His Ala Ile His Val Ser Gly Thr Asn Gly Thr Lys Arg Phe Asp
65 70 75 80
Asn Pro Val Leu Pro Phe Asn Asp Gly Val Tyr Phe Ala Ser Thr Glu
85 90 95
Lys Ser Asn Ile Ile Arg Gly Trp Ile Phe Gly Thr Thr Leu Asp Ser
100 105 110
Lys Thr Gln Ser Leu Leu Ile Val Asn Gln Ala Thr Asn Val Val Ile
115 120 125
Lys Val Cys Glu Phe Gln Phe Cys Asn Asp Pro Phe Leu Gly Val Tyr
130 135 140
Tyr His Lys Asn Asn Lys Ser Trp Met Glu Ser Glu Phe Arg Val Tyr
145 150 155 160
Ser Ser Ala Asn Gln Cys Thr Phe Glu Tyr Val Ser Gln Pro Phe Leu
165 170 175
Met Asp Leu Glu Gly Lys Gln Gly Asn Phe Lys Asn Leu Arg Glu Phe
180 185 190
Val Phe Lys Asn Ile Asp Gly Tyr Phe Lys Ile Tyr Ser Lys His Thr
195 200 205
Pro Ile Asn Leu Val Arg Asp Leu Pro Gln Gly Phe Ser Ala Leu Glu
210 215 220
Pro Leu Val Asp Leu Pro Ile Gly Ile Gln Ile Thr Arg Phe Gln Thr
225 230 235 240
Leu Leu Ala Leu His Arg Ser Tyr Leu Thr Pro Gly Asp Ser Ser Ser
245 250 255
Gly Trp Thr Ala Gly Ala Ala Ala Tyr Tyr Val Gly Tyr Leu Gln Pro
260 265 270
Arg Thr Phe Leu Leu Lys Tyr Asn Glu Asn Gly Thr Ile Thr Asp Ala
275 280 285
Val Asp Cys Ala Leu Asp Pro Leu Ser Glu Thr Lys Cys Thr Leu Lys
290 295 300
Ser Phe Thr Val Glu Lys Gly Ile Tyr Gln Thr Ser Asn Phe Arg Val
305 310 315 320
Gln Pro Ala Glu Ala Ile Val Arg Phe Pro Gln Ile Thr Asn Leu Cys
325 330 335
Pro Phe Gly Glu Val Phe Gln Ala Thr Arg Phe Ala Ser Val Tyr Ala
340 345 350
Trp Asn Arg Lys Arg Ile Ser Asn Cys Val Ala Asp Tyr Ser Val Leu
355 360 365
Tyr Asn Ser Ala Ser Phe Ser Thr Phe Lys Cys Tyr Gly Val Ser Pro
370 375 380
Thr Lys Leu Asn Asp Leu Cys Phe Thr Asn Val Tyr Ala Asp Ser Phe
385 390 395 400
Val Ile Arg Gly Asp Glu Val Arg Gln Ile Ala Pro Gly Gln Thr Gly
405 410 415
Lys Ile Ala Asp Tyr Asn Tyr Lys Leu Pro Asp Asp Phe Thr Gly Cys
420 425 430
Val Ile Ala Trp Asn Ser Asn Asn Leu Asp Ser Lys Val Gly Gly Asn
435 440 445
Tyr Asn Tyr Leu Tyr Arg Leu Phe Arg Lys Ser Asn Leu Lys Pro Phe
450 455 460
Glu Arg Asp Ile Ser Thr Glu Ile Tyr Gln Ala Gly Ser Thr Pro Cys
465 470 475 480
Asn Gly Val Glu Gly Phe Asn Cys Tyr Phe Pro Leu Gln Ser Tyr Gly
485 490 495
Phe Gln Pro Thr Asn Gly Val Gly Tyr Gln Pro Tyr Arg Val Val Val
500 505 510
Leu Ser Phe Glu Leu Leu His Ala Pro Ala Thr Val Cys Gly Pro Lys
515 520 525
Lys Ser Thr Asn Leu Val Lys Asn Lys Cys Val Asn Phe Asn Phe Asn
530 535 540
Gly Leu Thr Gly Thr Gly Val Leu Thr Glu Ser Asn Lys Lys Phe Leu
545 550 555 560
Pro Phe Gln Gln Phe Gly Arg Asp Ile Ala Asp Thr Thr Asp Ala Val
565 570 575
Arg Asp Pro Gln Thr Leu Glu Ile Leu Asp Ile Thr Pro Cys Ser Phe
580 585 590
Gly Gly Val Ser Val Ile Thr Pro Gly Thr Asn Thr Ser Asn Gln Val
595 600 605
Ala Val Leu Tyr Gln Asp Val Asn Cys Thr Glu Val Pro Val Ala Ile
610 615 620
His Ala Asp Gln Leu Thr Pro Thr Trp Arg Val Tyr Ser Thr Gly Ser
625 630 635 640
Asn Val Phe Gln Thr Arg Ala Gly Cys Leu Ile Gly Ala Glu His Val
645 650 655
Asn Asn Ser Tyr Glu Cys Asp Ile Pro Ile Gly Ala Gly Ile Cys Ala
660 665 670
Ser Tyr Gln Thr Gln Thr Asn Ser Pro Arg Arg Ala Arg Ser Val Ala
675 680 685
Ser Gln Ser Ile Ile Ala Tyr Thr Met Ser Leu Gly Ala Glu Asn Ser
690 695 700
Val Ala Tyr Ser Asn Asn Ser Ile Ala Ile Pro Thr Asn Phe Thr Ile
705 710 715 720
Ser Val Thr Thr Glu Ile Leu Pro Val Ser Met Thr Lys Thr Ser Val
725 730 735
Asp Cys Thr Met Tyr Ile Cys Gly Asp Ser Thr Glu Cys Ser Asn Leu
740 745 750
Leu Leu Gln Tyr Gly Ser Phe Cys Thr Gln Leu Asn Arg Ala Leu Thr
755 760 765
Gly Ile Ala Val Glu Gln Asp Lys Asn Thr Gln Glu Val Phe Ala Gln
770 775 780
Val Lys Gln Ile Tyr Lys Thr Pro Pro Ile Lys Asp Phe Gly Gly Phe
785 790 795 800
Asn Phe Ser Gln Ile Leu Pro Asp Pro Ser Lys Pro Ser Lys Arg Ser
805 810 815
Phe Ile Glu Asp Leu Leu Phe Asn Lys Val Thr Leu Ala Asp Ala Gly
820 825 830
Phe Ile Lys Gln Tyr Gly Asp Cys Leu Gly Asp Ile Ala Ala Arg Asp
835 840 845
Leu Ile Cys Ala Gln Lys Phe Asn Gly Leu Thr Val Leu Pro Pro Leu
850 855 860
Leu Thr Asp Glu Met Ile Ala Gln Tyr Thr Ser Ala Leu Leu Ala Gly
865 870 875 880
Thr Ile Thr Ser Gly Trp Thr Phe Gly Ala Gly Ala Ala Leu Gln Ile
885 890 895
Pro Phe Ala Met Gln Met Ala Tyr Arg Phe Asn Gly Ile Gly Val Thr
900 905 910
Gln Asn Val Leu Tyr Glu Asn Gln Lys Leu Ile Ala Asn Gln Phe Asn
915 920 925
Ser Ala Ile Gly Lys Ile Gln Asp Ser Leu Ser Ser Thr Ala Ser Ala
930 935 940
Leu Gly Lys Leu Gln Asp Val Val Asn Gln Asn Ala Gln Ala Leu Asn
945 950 955 960
Thr Leu Val Lys Gln Leu Ser Ser Asn Phe Gly Ala Ile Ser Ser Val
965 970 975
Leu Asn Asp Ile Leu Ser Arg Leu Asp Pro Pro Glu Ala Glu Val Gln
980 985 990
Ile Asp Arg Leu Ile Thr Gly Arg Leu Gln Ser Leu Gln Thr Tyr Val
995 1000 1005
Thr Gln Gln Leu Ile Arg Ala Ala Glu Ile Arg Ala Ser Ala Asn
1010 1015 1020
Leu Ala Ala Thr Lys Met Ser Glu Cys Val Leu Gly Gln Ser Lys
1025 1030 1035
Arg Val Asp Phe Cys Gly Lys Gly Tyr His Leu Met Ser Phe Pro
1040 1045 1050
Gln Ser Ala Pro His Gly Val Val Phe Leu His Val Thr Tyr Val
1055 1060 1065
Pro Ala Gln Glu Lys Asn Phe Thr Thr Ala Pro Ala Ile Cys His
1070 1075 1080
Asp Gly Lys Ala His Phe Pro Arg Glu Gly Val Phe Val Ser Asn
1085 1090 1095
Gly Thr His Trp Phe Val Thr Gln Arg Asn Phe Tyr Glu Pro Gln
1100 1105 1110
Ile Ile Thr Thr Asp Asn Thr Phe Val Ser Gly Asn Cys Asp Val
1115 1120 1125
Val Ile Gly Ile Val Asn Asn Thr Val Tyr Asp Pro Leu Gln Pro
1130 1135 1140
Glu Leu Asp Ser Phe Lys Glu Glu Leu Asp Lys Tyr Phe Lys Asn
1145 1150 1155
His Thr Ser Pro Asp Val Asp Leu Gly Asp Ile Ser Gly Ile Asn
1160 1165 1170
Ala Ser Val Val Asn Ile Gln Lys Glu Ile Asp Arg Leu Asn Glu
1175 1180 1185
Val Ala Lys Asn Leu Asn Glu Ser Leu Ile Asp Leu Gln Glu Leu
1190 1195 1200
Gly Lys Tyr Glu Gln Tyr Ile Lys Trp Pro Trp Tyr Ile Trp Leu
1205 1210 1215
Gly Phe Ile Ala Gly Leu Ile Ala Ile Val Met Val Thr Ile Met
1220 1225 1230
Leu Cys Cys Met Thr Ser Cys Cys Ser Cys Leu Lys Gly Cys Cys
1235 1240 1245
Ser Cys Gly Ser Cys Cys Lys Phe Asp Glu Asp Asp Ser Glu Pro
1250 1255 1260
Val Leu Lys Gly Val Lys Leu His Tyr Thr
1265 1270
<210> 41
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> immunogenic peptide
<400> 41
Thr Glu Ser Ile Val Arg Phe Pro Asn Ile Thr Asn Leu
1 5 10
<210> 42
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> immunogenic peptide
<400> 42
Asn Ile Thr Asn Leu Cys Pro Phe Gly Glu Val Phe Asn Ala Thr Arg
1 5 10 15
<210> 43
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> immunogenic peptide
<400> 43
Leu Tyr Asn Ser Ala Ser Phe Ser Thr Phe Lys
1 5 10
<210> 44
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> immunogenic peptide
<400> 44
Leu Asp Ser Lys Val Gly Gly Asn Tyr Asn
1 5 10
<210> 45
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> immunogenic peptide
<400> 45
Lys Ser Asn Leu Lys Pro Phe Glu Arg Asp Ile Ser Thr
1 5 10
<210> 46
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> immunogenic peptide
<400> 46
Lys Pro Phe Glu Arg Asp Ile Ser Thr Glu Ile Tyr Gln Ala Gly
1 5 10 15
<210> 47
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> immunogenic peptide
<400> 47
Gly Pro Lys Lys Ser Thr Asn Leu Val Lys Asn Lys Cys
1 5 10
<210> 48
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> immunogenic peptide
<400> 48
Asn Cys Asp Val Val Ile Gly Ile Val Asn Asn Thr Val Tyr
1 5 10
<210> 49
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<223> immunogenic peptide
<400> 49
Pro Glu Leu Asp Ser Phe Lys Glu Glu Leu Asp Lys Tyr Phe Lys Asn
1 5 10 15
His Thr Ser
<210> 50
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> immunogenic peptide
<400> 50
Val Asn Ile Gln Lys Glu Ile Asp Arg Leu Asn Glu Val Ala
1 5 10
<210> 51
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> immunogenic peptide
<400> 51
Asn Leu Asn Glu Ser Leu Ile Asp Leu Gln
1 5 10
<210> 52
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> immunogenic peptide
<400> 52
Leu Gly Lys Tyr Glu Gln Tyr Ile Lys Trp Pro
1 5 10
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Вакцина на основе AAV5 для индукции специфического иммунитета к вирусу SARS-CoV-2 и/или профилактики коронавирусной инфекции, вызванной SARS-CoV-2 | 2020 |
|
RU2783313C1 |
| Вакцина на основе AAV5 для индукции специфического иммунитета к вирусу SARS-CoV-2 и/или профилактики коронавирусной инфекции, вызванной SARS-CoV-2 | 2021 |
|
RU2761879C1 |
| Вакцина на основе AAV5 для индукции специфического иммунитета к вирусу SARS-CoV-2 и/или профилактики коронавирусной инфекции, вызванной SARS-CoV-2 | 2020 |
|
RU2760301C1 |
| УСОВЕРШЕНСТВОВАННАЯ КОРОНАВИРУСНАЯ ВАКЦИНА | 2022 |
|
RU2816182C2 |
| АНТИТЕЛА ПРОТИВ SARS-COV-2 И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2021 |
|
RU2832025C1 |
| БЕЛОК И ВАКЦИНА ПРОТИВ ИНФЕКЦИИ SARS-CoV-2 | 2020 |
|
RU2815060C1 |
| СПОСОБ ПОВЫШЕНИЯ ИММУНОГЕННОСТИ БЕЛКОВОГО/ПЕПТИДНОГО АНТИГЕНА | 2021 |
|
RU2830627C1 |
| Генная конструкция для экспрессии рекомбинантных белков на основе участка S-белка SARS-CoV-2, включающего RBD и SD1, слитого с Fc фрагментом IgG, способ получения рекомбинантных белков, антигены и антигенные композиции для индукции длительного антительного и клеточного иммунитета против вируса SARS-CoV-2 | 2021 |
|
RU2802825C2 |
| Однодоменное антитело и его модификации, специфически связывающиеся с RBD S белка вируса SARS-CoV-2, и способ их применения для терапии и экстренной профилактики заболеваний, вызываемых вирусом SARS-CoV-2 | 2021 |
|
RU2763001C1 |
| СЛИТЫЙ ПОЛИПЕПТИД, ВКЛЮЧАЮЩИЙ GDF15 И ПОЛИПЕПТИДНУЮ ОБЛАСТЬ, СПОСОБНУЮ К О-ГЛИКОЗИЛИРОВАНИЮ | 2020 |
|
RU2812049C1 |


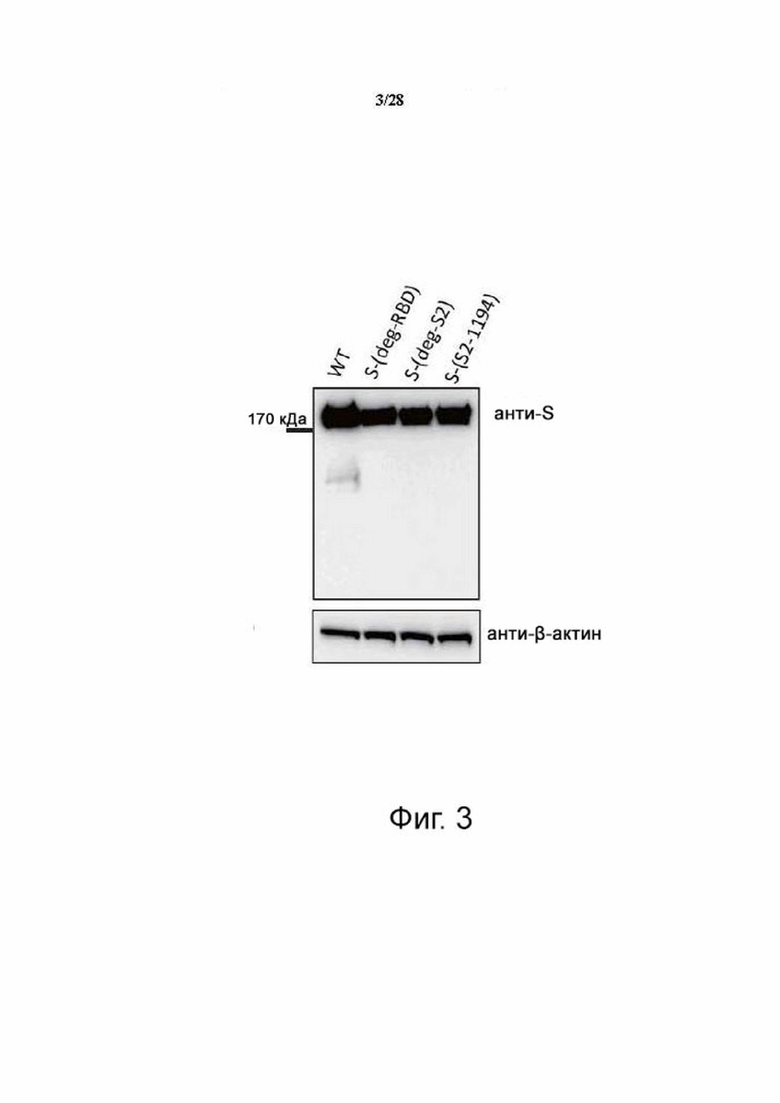
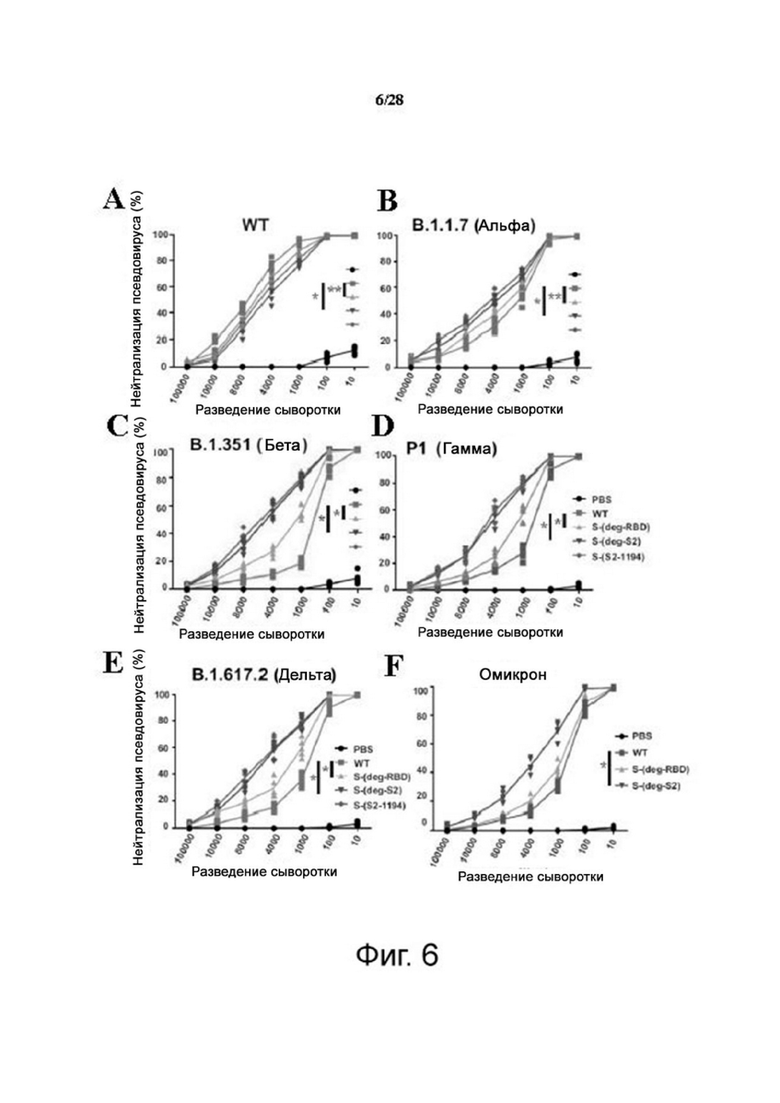
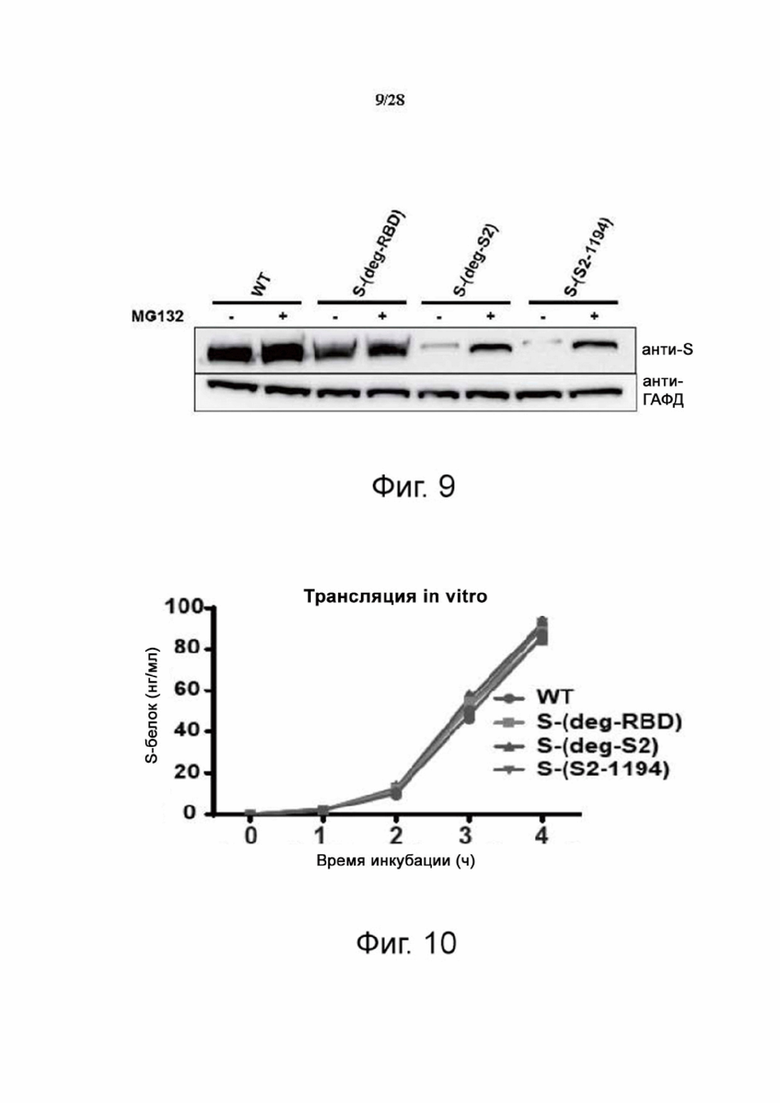
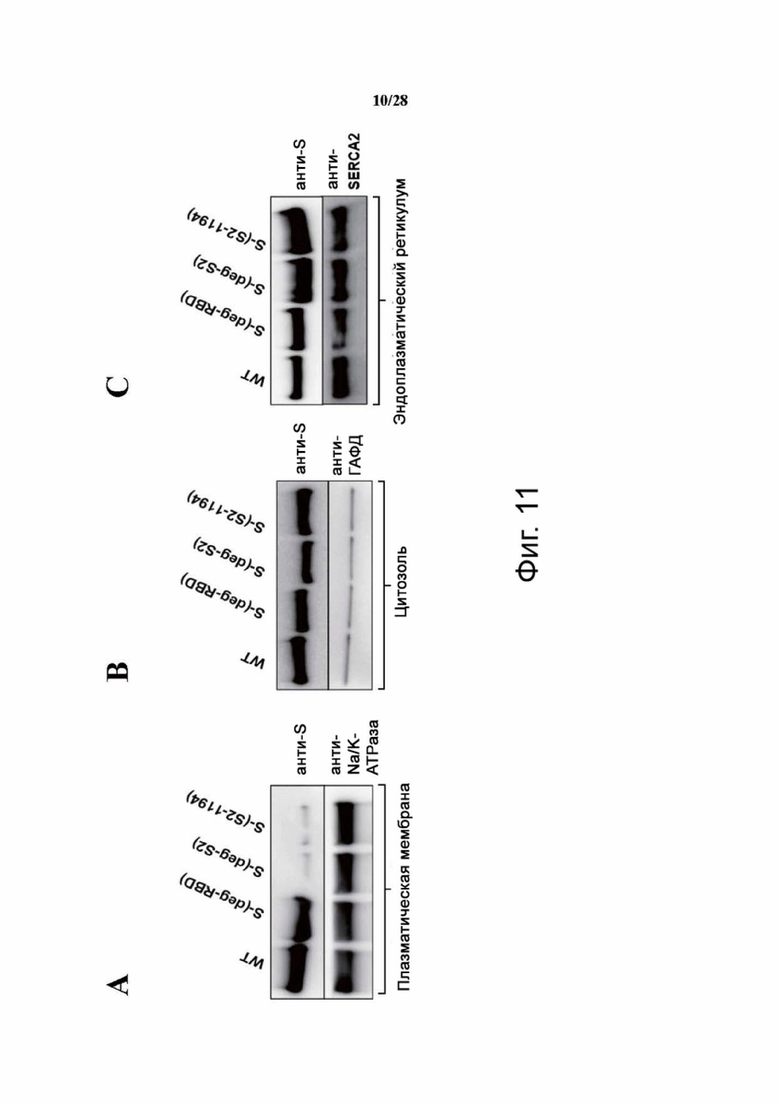
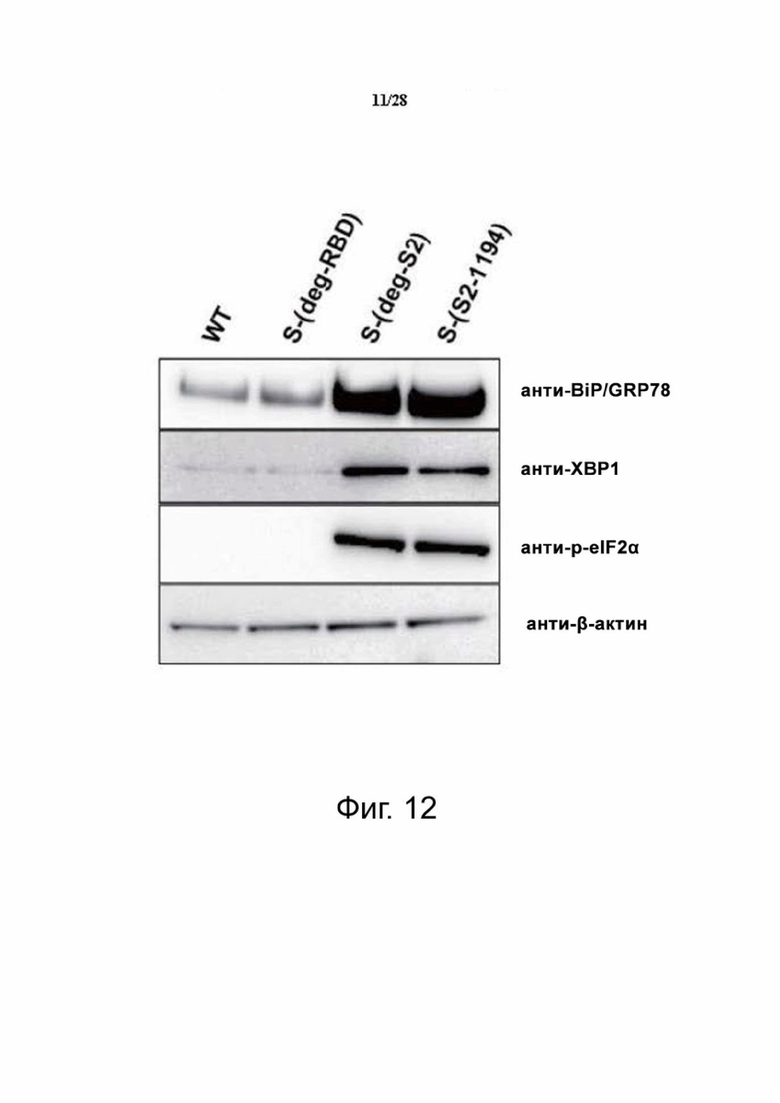
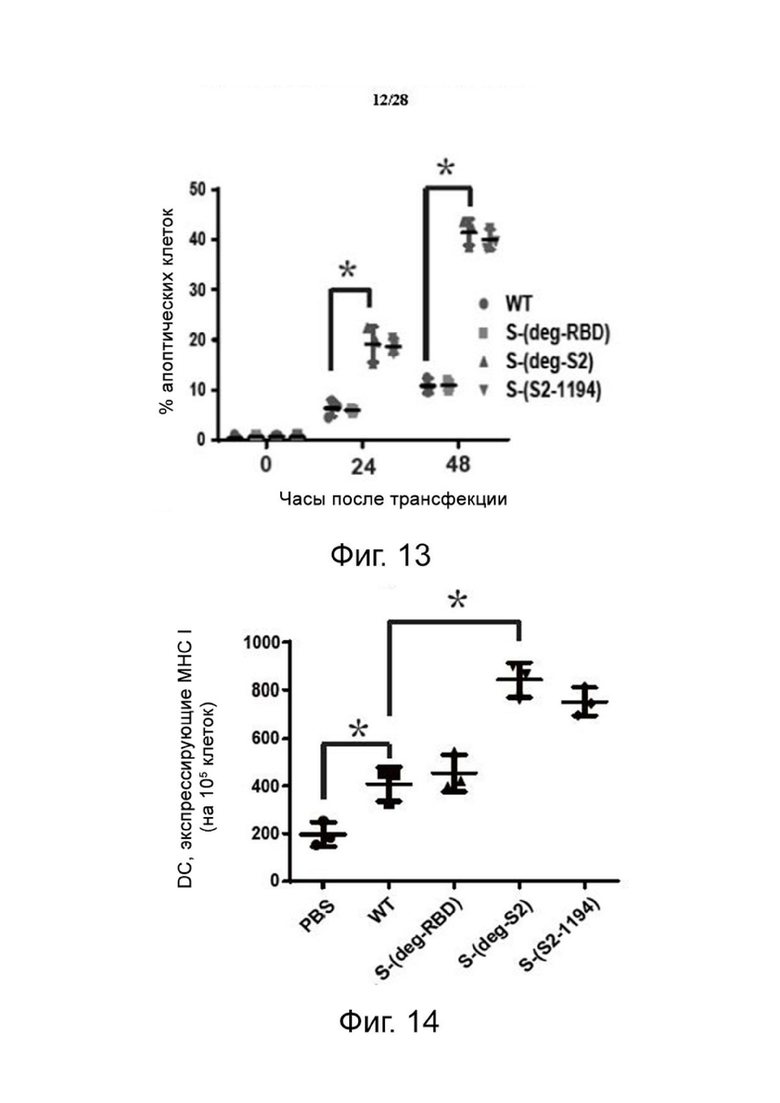
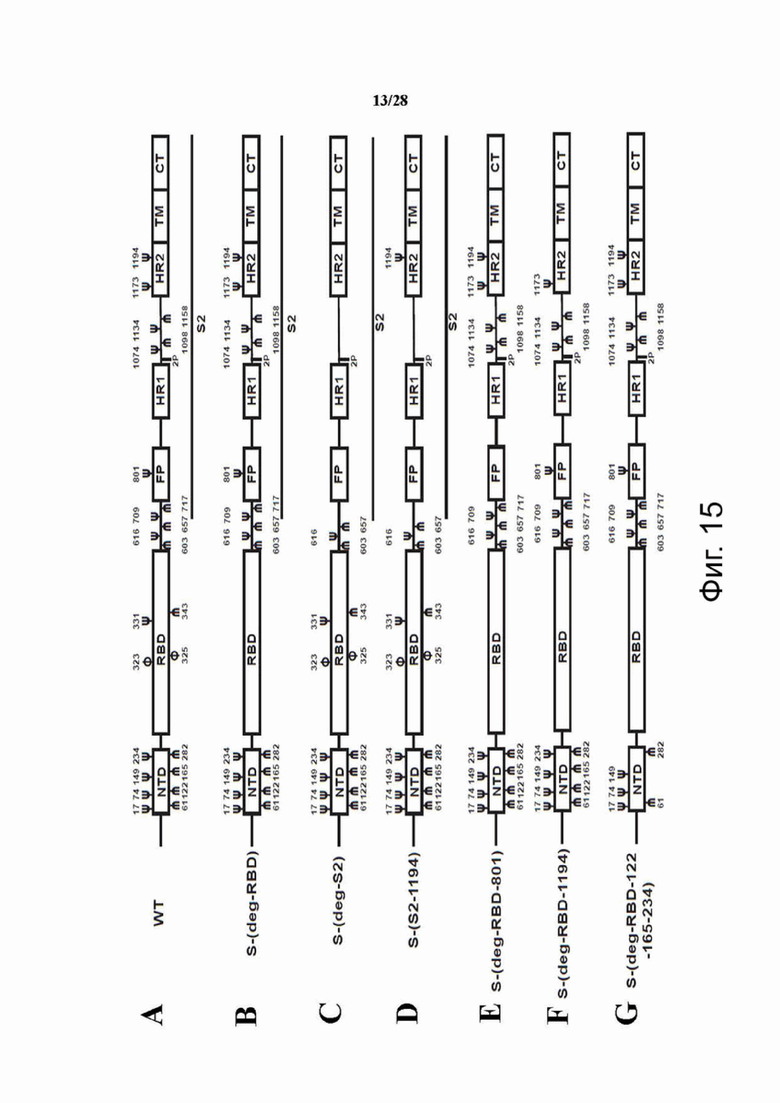
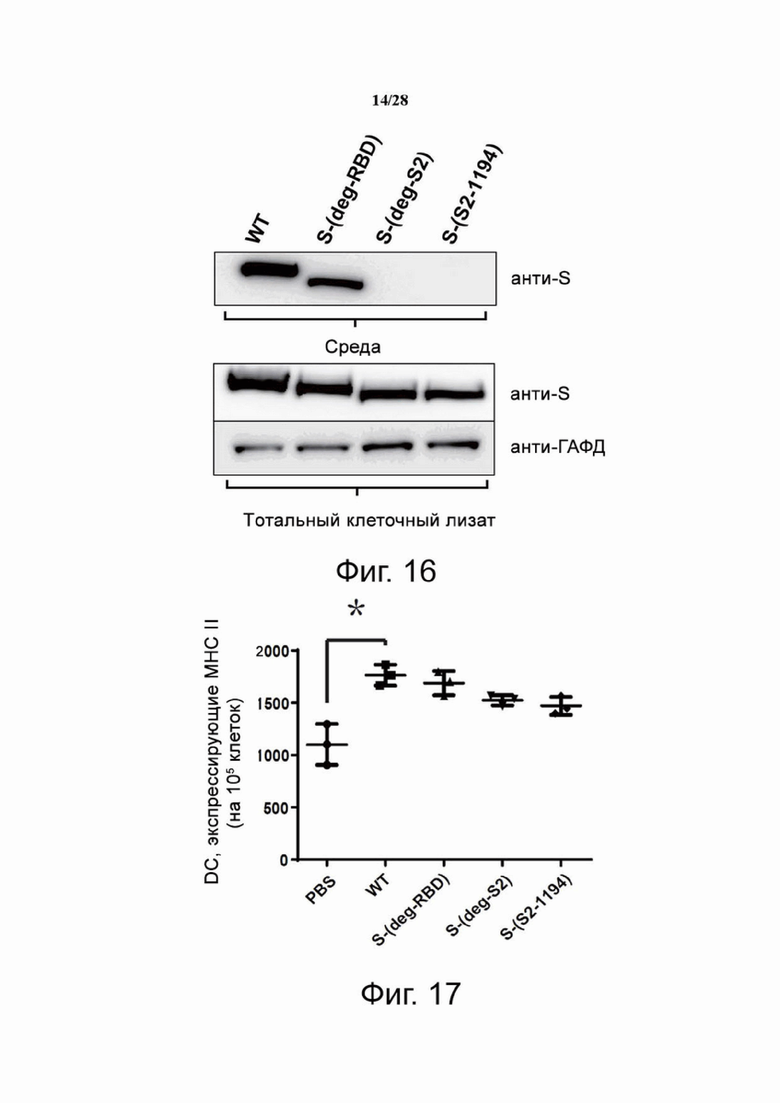
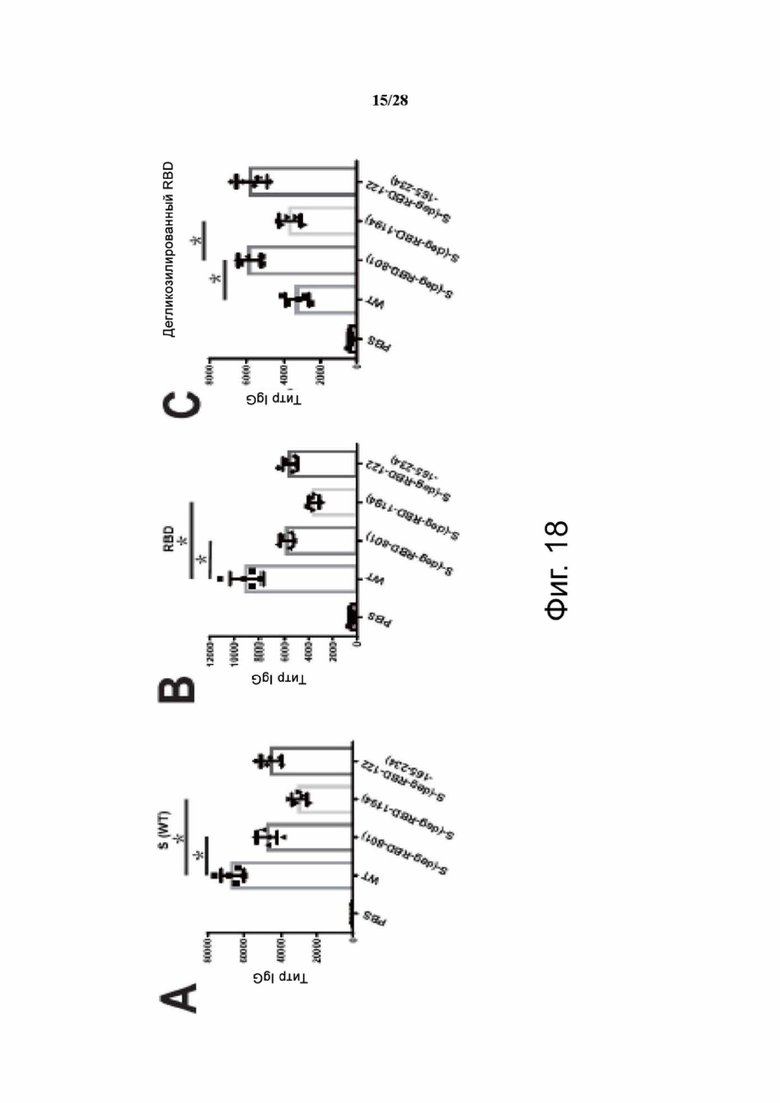
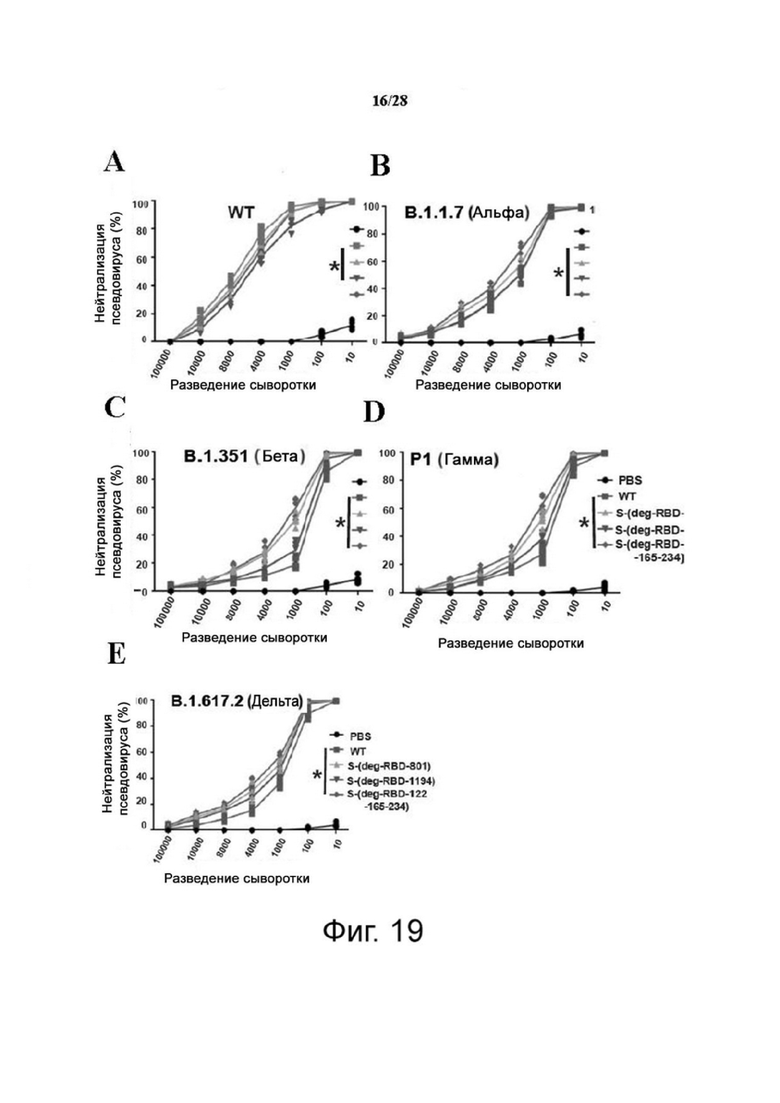
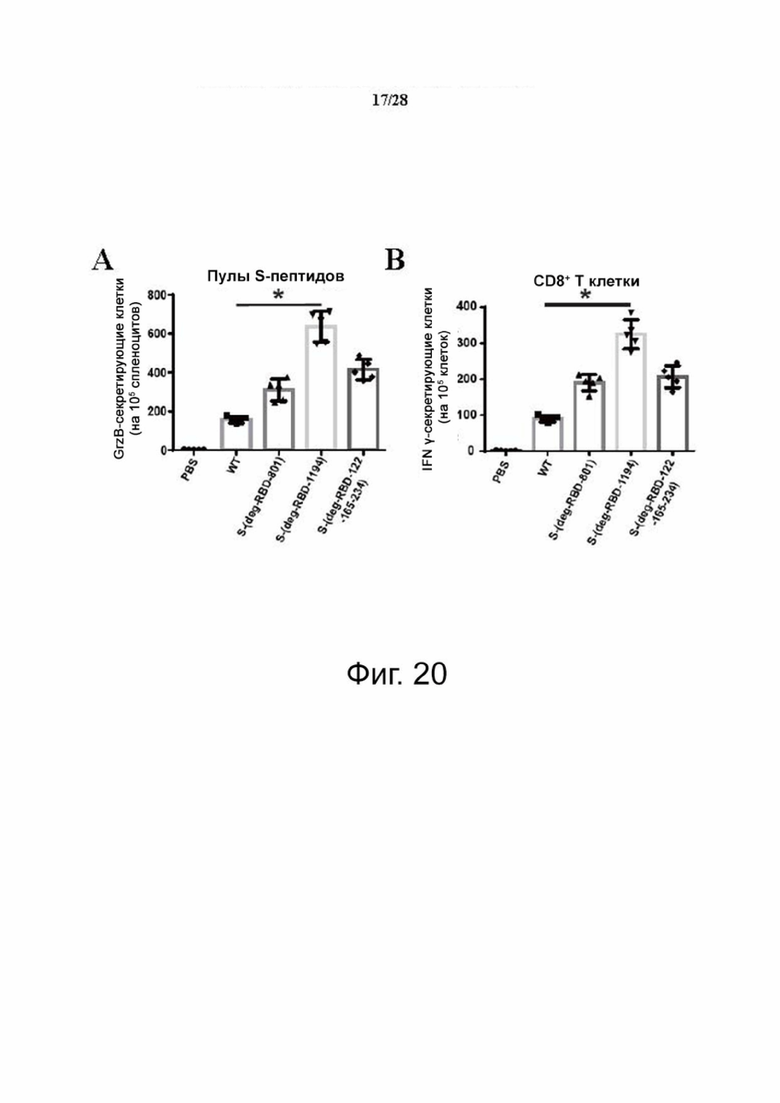
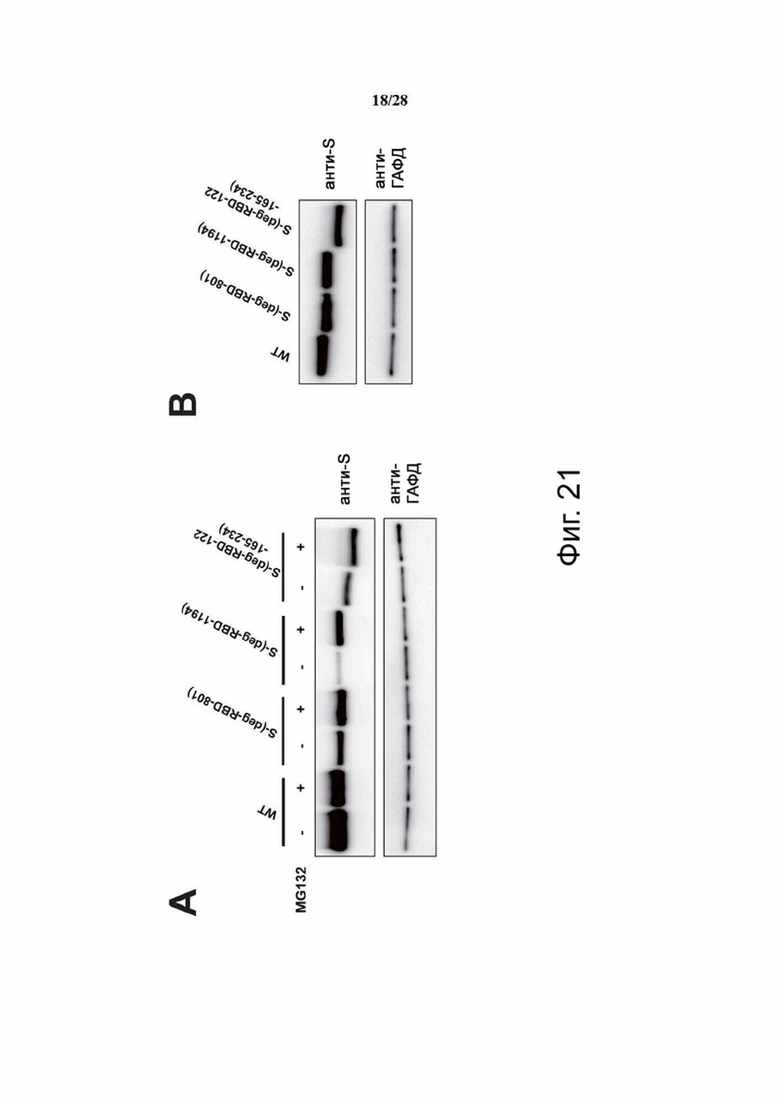
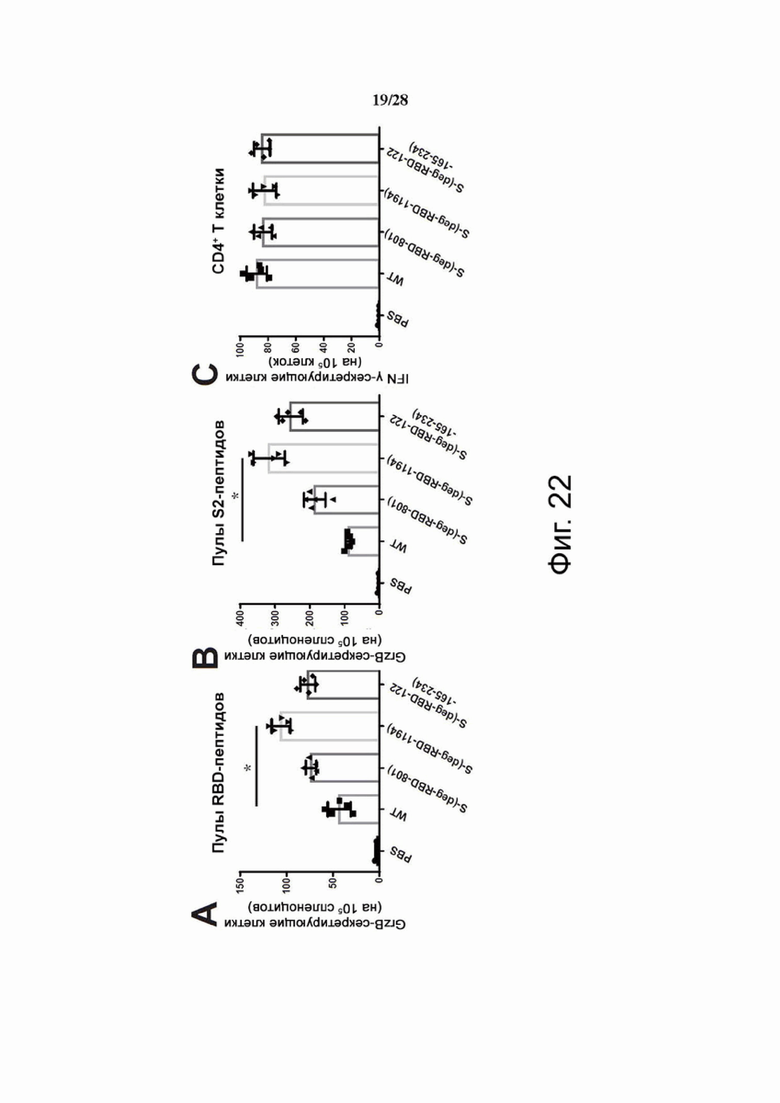
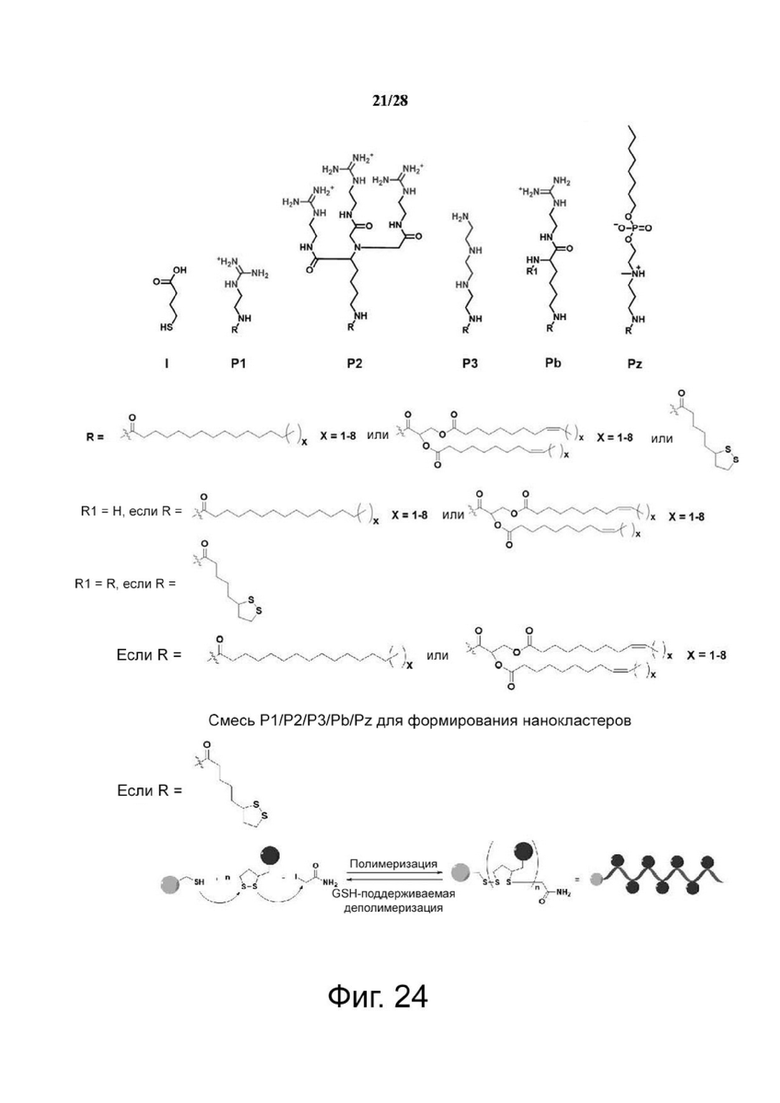
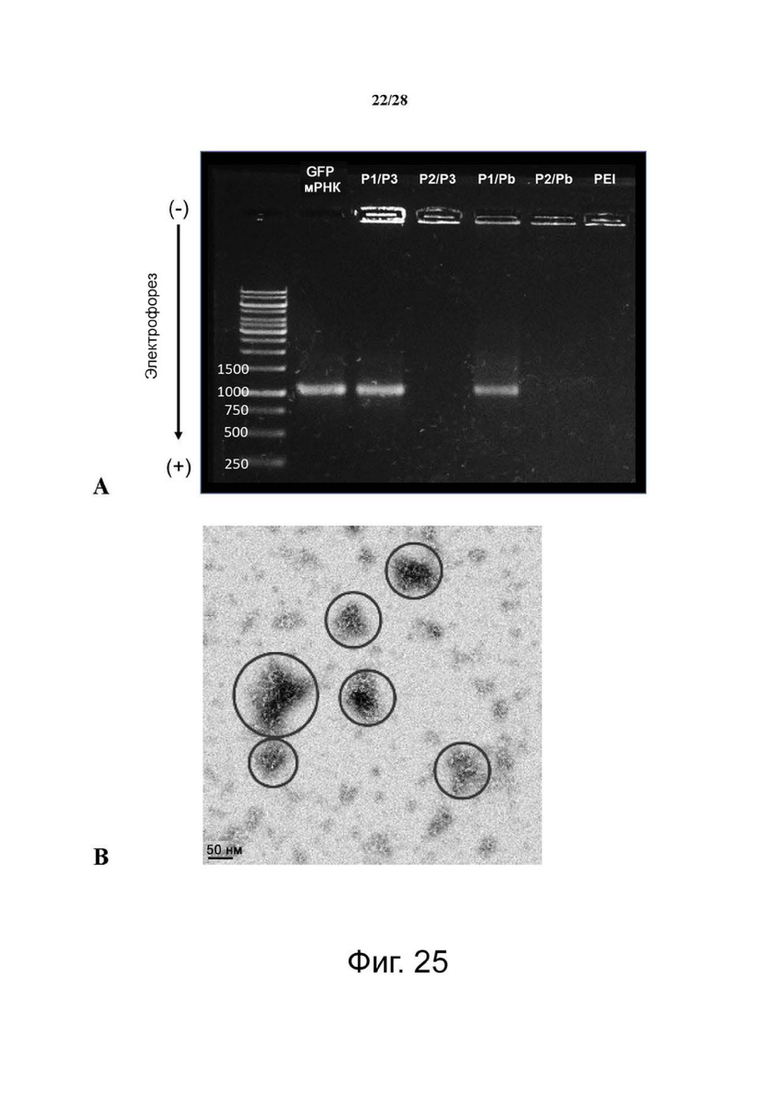

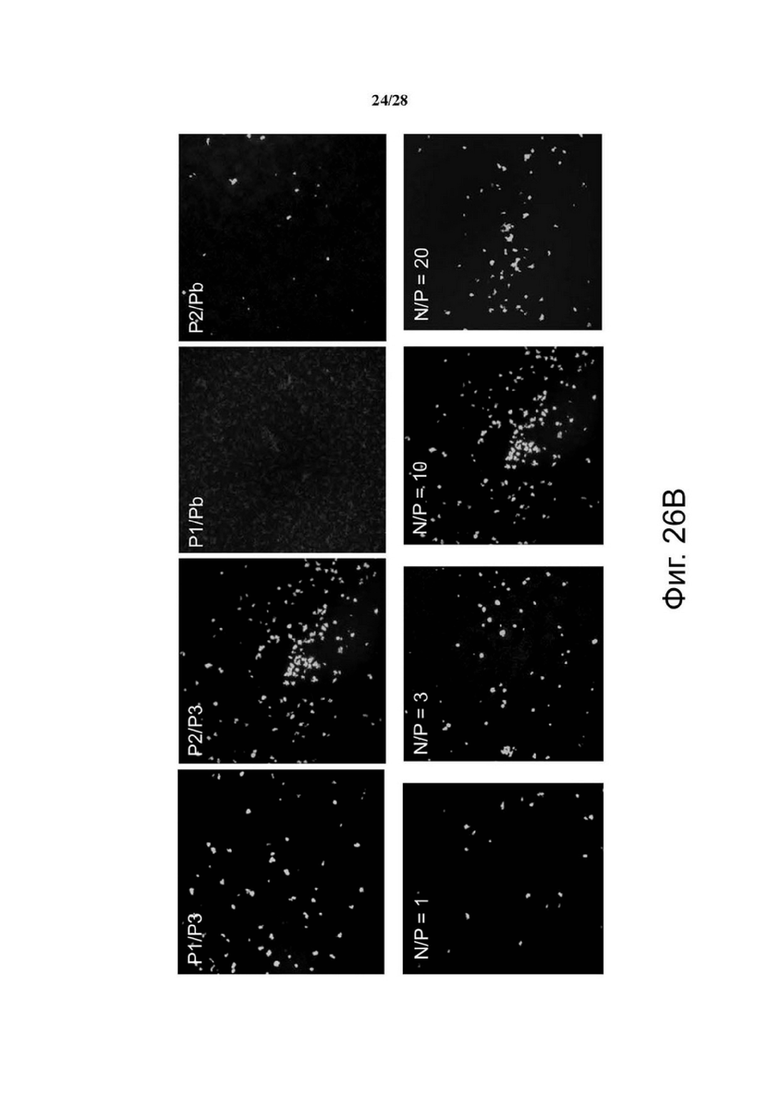

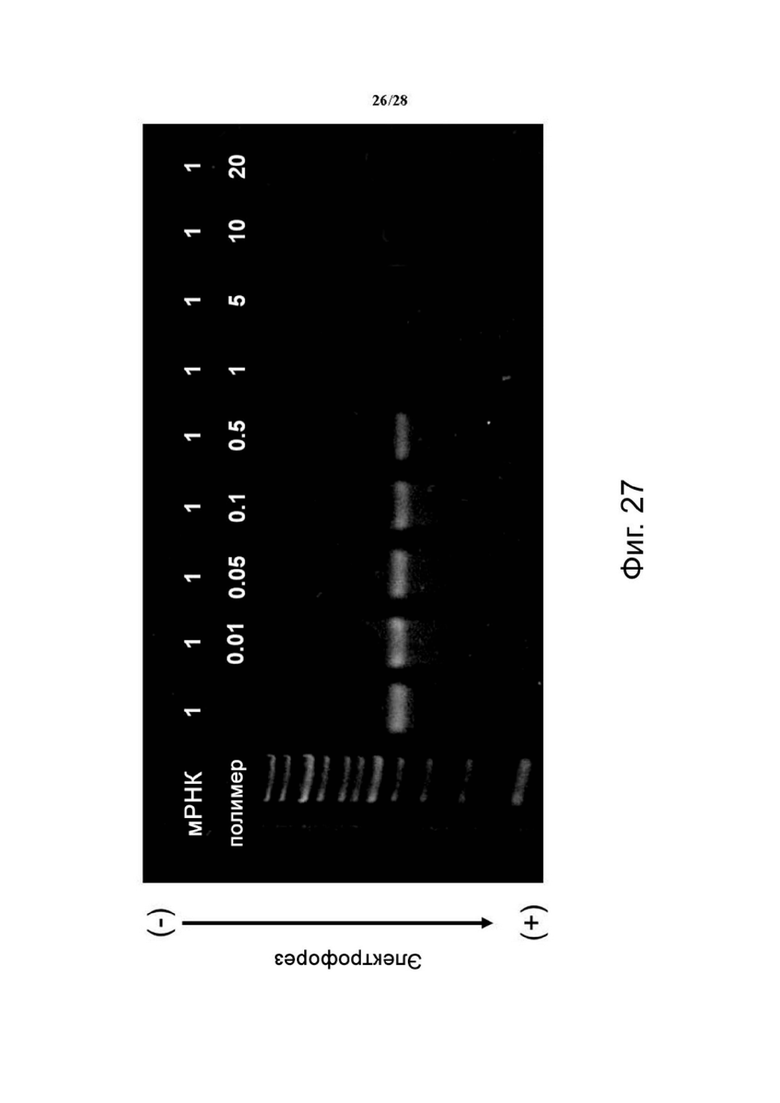

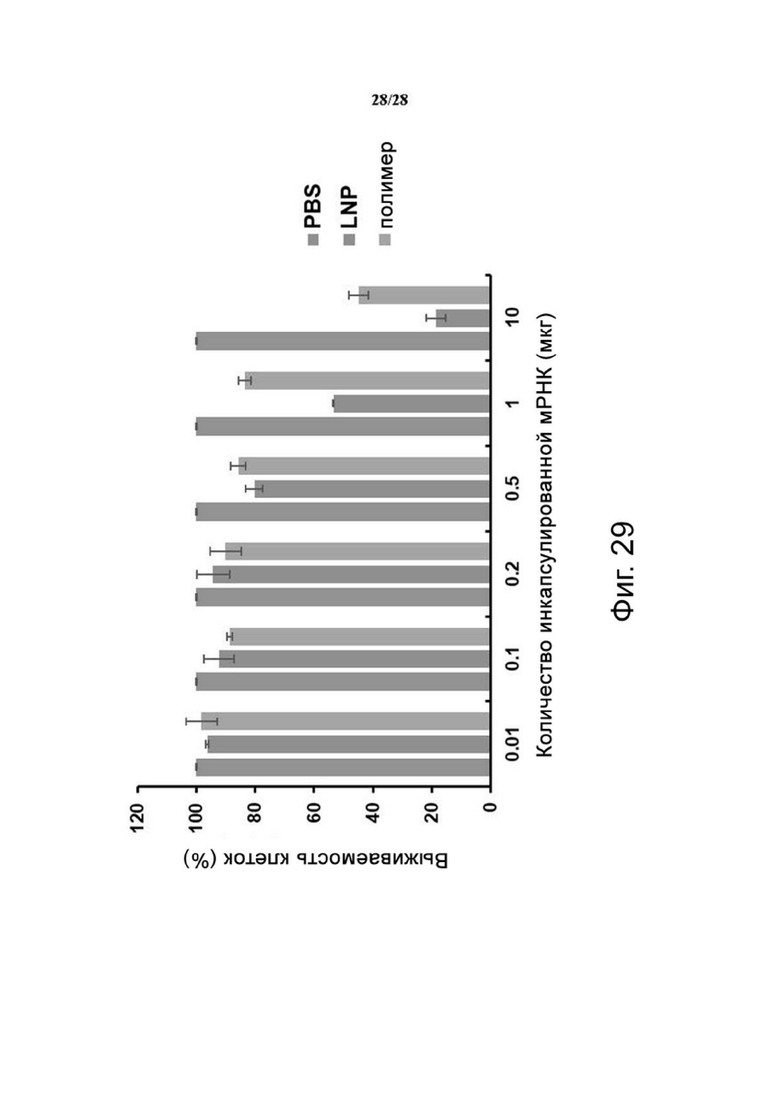
Изобретение относится к биотехнологии. Описана молекула модифицированной нуклеиновой кислоты, кодирующая модифицированный шиповидный белок, полученный из шиповидного белка SARS-CoV-2, имеющего делецию сайтов гликозилирования в рецептор-связывающем домене (RBD), домене субъединицы 1 (S1) или домене субъединицы 2 (S2). Также описан способ профилактики или лечения коронавирусной инфекции, включающий введение композиции с эффективным количеством модифицированной молекулы нуклеиновой кислоты субъекту, инфицированному или подверженному риску заражения коронавирусом. Вакцина вызывает широкий защитный иммунный ответ против коронавируса и его вариантов. 2 н. и 17 з.п. ф-лы, 29 ил., 2 табл., 1 пр.
1. Молекула модифицированной нуклеиновой кислоты, кодирующая модифицированный шиповидный белок, полученный из шиповидного белка SARS-CoV-2, имеющего делецию сайтов гликозилирования в рецептор-связывающем домене (RBD), домене субъединицы 1 (S1) или домене субъединицы 2 (S2);
где в указанном модифицированном шиповидном белке все 2 N-сайты гликозилирования в RBD мутированы с N на Q, а 2 O-сайты гликозилирования мутированы с S/T на A;
где в указанном модифицированном шиповидном белке все 8 сайтов гликозилирования в S2 мутированы с N на Q, кроме сайта гликозилизования 1194, где модифицированный шиповидный белок содержит аминокислотную последовательность согласно SEQ ID NO: 2 или 18; или
где в указанном модифицированном шиповидном белке один или несколько сайтов гликозилирования 801, 1194, 122, 165 и 234 в S2 мутированы с N на Q, где модифицированный шиповидный белок содержит аминокислотную последовательность согласно SEQ ID NO: 2 или 18;
где указанный модифицированный шиповидный белок содержит аминокислотную последовательность согласно SEQ ID NO: 2, 16, 18 или 20, или аминокислотную последовательность, имеющую по меньшей мере примерно 95% идентичности с аминокислотной последовательностью SEQ ID NO: 2, 16, 18 или 20;
где модифицированная молекула нуклеиновой кислоты представляет собой мРНК или двухцепочечную или одноцепочечную ДНК.
2. Молекула модифицированной нуклеиновой кислоты по п. 1, в которой модифицированный шиповидный белок содержит аминокислотную последовательность SEQ ID NO: 2, 16, 18 или 20 или аминокислотную последовательность, имеющую, по меньшей мере, примерно 99%, 98%, 97% или 96% идентичности с аминокислотной последовательностью SEQ ID NO: 2, 16, 18 или 20.
3. Молекула модифицированной нуклеиновой кислоты по п. 2, в которой молекула нуклеиновой кислоты, кодирующая аминокислотную последовательность SEQ ID NO: 2, 16, 18 или 20, представляет собой мРНК, содержащую нуклеотидную последовательность SEQ ID NO: 1, 15, 17 или 19, соответственно, или нуклеотидную последовательность, имеющую, по меньшей мере, примерно 99%, 98%, 97%, 96%, 95%, 90%, 85% или 80% идентичности с нуклеотидной последовательностью SEQ ID NO: 1, 15, 17 или 19, соответственно.
4. Молекула модифицированной нуклеиновой кислоты по п. 1, в которой модифицированный шиповидный белок содержит аминокислотную последовательность SEQ ID NO: 4, 22, 24 или 26, при этом модифицированный шиповидный белок содержит рецептор-связывающий домен (RBD), в котором отсутствуют сайты гликозилирования.
5. Молекула модифицированной нуклеиновой кислоты по п. 4, в которой молекула модифицированной нуклеиновой кислоты, кодирующая аминокислотную последовательность SEQ ID NO: 4, 22, 24 или 26, содержит нуклеотидную последовательность SEQ ID NO: 3, 21, 23 или 25, соответственно, или нуклеотидную последовательность, по меньшей мере, примерно 99%, 98%, 97%, 96%, 95%, 90%, 85% или 80% идентичности с нуклеотидной последовательностью SEQ ID NO: 3, 21, 23 или 25, соответственно.
6. Молекула модифицированной нуклеиновой кислоты по п. 1, в которой модифицированный шиповидный белок содержит аминокислотную последовательность SEQ ID NO: 6, 28, 30 или 32, в которой модифицированный шиповидный белок содержит субъединицу S2, лишенную сайтов гликозилирования.
7. Молекула модифицированной нуклеиновой кислоты по п. 6, в которой молекула модифицированной нуклеиновой кислоты, кодирующая аминокислотную последовательность SEQ ID NO: 6, 28, 30 или 32, включает нуклеотидную последовательность SEQ ID NO: 5, 27, 29 или 31, соответственно, или нуклеотидную последовательность, имеющую, по меньшей мере, примерно 99%, 98%, 97%, 96%, 95%, 90%, 85% или 80% идентичности с нуклеотидной последовательностью SEQ ID NO: 5, 27, 29 или 31, соответственно.
8. Молекула модифицированной нуклеиновой кислоты по п. 1, в которой модифицированный шиповидный белок, описанный в настоящем документе, содержит аминокислотную последовательность SEQ ID NO: 8 или 34, в которой модифицированный шиповидный белок содержит субъединицу S2, содержащую единственный сайт гликозилирования, где единственный сайт гликозилирования находится в положении N1194.
9. Молекула модифицированной нуклеиновой кислоты по п. 8, в которой молекула модифицированной нуклеиновой кислоты, кодирующая аминокислотную последовательность SEQ ID NO: 8 или 34, содержит нуклеотидную последовательность SEQ ID NO: 7 или 33, соответственно, или нуклеотидную последовательность, имеющую, по меньшей мере, примерно 99%, 98%, 97%, 96%, 95%, 90%, 85% или 80% идентичности с нуклеотидной последовательностью SEQ ID NO: 7 или 33, соответственно.
10. Молекула модифицированной нуклеиновой кислоты по п. 1, в которой модифицированный шиповидный белок содержит аминокислотную последовательность SEQ ID NO: 10 или 36, при этом модифицированный шиповидный белок содержит рецептор-связывающий домен (RBD), в котором отсутствуют сайты гликозилирования, и аминокислотную замену N801 на Q801.
11. Молекула модифицированной нуклеиновой кислоты по п. 10, в которой молекула модифицированной нуклеиновой кислоты, кодирующая аминокислотную последовательность SEQ ID NO: 10 или 36, содержит нуклеотидную последовательность SEQ ID NO: 9 или 35, соответственно, или нуклеотидную последовательность, имеющую, по меньшей мере, примерно 99%, 98%, 97%, 96%, 95%, 90%, 85% или 80% идентичности с нуклеотидной последовательностью SEQ ID NO: 9 или 35, соответственно.
12. Молекула модифицированной нуклеиновой кислоты по п. 1, в которой модифицированный шиповидный белок содержит аминокислотную последовательность SEQ ID NO: 12 или 38, при этом модифицированный шиповидный белок содержит рецептор-связывающий домен (RBD), в котором отсутствуют сайты гликозилирования, и аминокислотную замену N1194 на Q1194.
13. Молекула модифицированной нуклеиновой кислоты по п. 12, в которой молекула модифицированной нуклеиновой кислоты, кодирующая аминокислотную последовательность SEQ ID NO: 12 или 38, содержит нуклеотидную последовательность SEQ ID NO: 11 или 37, соответственно, или нуклеотидную последовательность, имеющую, по меньшей мере, примерно 99%, 98%, 97%, 96%, 95%, 90%, 85% или 80% идентичности с нуклеотидной последовательностью SEQ ID NO: 11 или 37, соответственно.
14. Молекула модифицированной нуклеиновой кислоты по п. 1, в которой модифицированный шиповидный белок, описанный в настоящем документе, содержит аминокислотную последовательность SEQ ID NO: 14 или 40, при этом модифицированный шиповидный белок содержит модифицированный рецептор-связывающий домен (RBD), в котором отсутствуют сайты гликозилирования, и аминокислотные замены N122 на Q122, N165 на Q165 и N234 на Q234.
15. Молекула модифицированной нуклеиновой кислоты по п. 14, в которой молекула модифицированной нуклеиновой кислоты, кодирующая аминокислотную последовательность SEQ ID NO: 14 или 40, содержит нуклеотидную последовательность SEQ ID NO: 13 или 39, соответственно, или нуклеотидную последовательность, имеющую, по меньшей мере, примерно 99%, 98%, 97%, 96%, 95%, 90%, 85% или 80% идентичности с нуклеотидной последовательностью SEQ ID NO: 13 или 39, соответственно.
16. Молекула модифицированной нуклеиновой кислоты по п. 1, в которой мРНК для S-(deg-RBD) имеет последовательность SEQ ID NO: 3, 21, 23 или 25, мРНК или ДНК для S-(deg-S2) имеет последовательность SEQ ID NO: 5, 27, 29 или 31, мРНК или ДНК для S-(deg-S2-1194) имеет последовательность SEQ ID NO: 7 или 33, мРНК или ДНК для S-(deg-RBD-801) имеет последовательность SEQ ID NO: 9 или 35, мРНК или ДНК для S-(deg-RBD-1194) имеет последовательность SEQ ID NO: 11 или 37, мРНК или ДНК для S-(deg-RBD-122-165-234) имеет последовательность SEQ ID NO: 13 или 39.
17. Молекула модифицированной нуклеиновой кислоты по любому из пп. 1-16, которую можно применять в качестве мРНК-вакцины против коронавируса.
18. Способ профилактики или лечения коронавирусной инфекции, включающий введение композиции с эффективным количеством молекулы модифицированной нуклеиновой кислоты по любому из пп. 1-17, субъекту, инфицированному или подверженному риску заражения коронавирусом.
19. Способ по п. 18, в котором вакцина сформулирована в виде комбинации, содержащей одну или несколько дополнительных вакцин, где каждый член комбинации содержит эффективное количество молекулы модифицированной нуклеиновой кислоты, в диапазоне от 5 мкг до 50 мкг.
| Jie Zheng, Yoshiyuki Yamada et al | |||
| Identification of N-linked glycosylation sites in the spike protein and their functional impact on the replication and infectivity of coronavirus infectious bronchitis virus in cell culture, Virology, 2018 Jan 1:513:65-74 | |||
| CN 112626124 A, 09.04.2021 | |||
| СПОСОБ ИСПОЛЬЗОВАНИЯ РЕКОМБИНАНТНЫХ БЕЛКОВ SARS-COV-2 В СОСТАВЕ ТЕСТ-СИСТЕМЫ ДЛЯ ИММУНОФЕРМЕНТНОГО АНАЛИЗА С ОПРЕДЕЛЕНИЕМ УРОВНЕЙ АНТИТЕЛ КЛАССОВ IgM, IgG, IgA В СЫВОРОТКЕ/ПЛАЗМЕ КРОВИ БОЛЬНЫХ COVID-19 | 2020 |
|
RU2730897C1 |
Авторы
Даты
2024-09-05—Публикация
2022-04-12—Подача