Сведения о перечне последовательностей
Перечень последовательностей, связанных с данной заявкой, представлен в текстовом формате вместо бумажной копии и включен в данное описание посредством ссылки. Имя текстового файла, содержащего список последовательностей: 930585_402WO _SEQUENCE_LISTING.txt. Текстовый файл - 328 КБ, был создан 24 февраля 2021 года и передается в электронном виде через EFS-Web.
Предшествующий уровень техники
В конце 2019 года в китайском городе Ухань (Wuhan) появился новый бетакоронавирус. По состоянию на 19 февраля 2021 года во всем мире было подтверждено около 110 миллионов случаев инфицирования этим вирусом (называемым, среди прочего, SARS-CoV-2 (коронавирус-2 тяжелого острого респираторного синдрома) и первоначально идентифицированным как коронавирус Ухань), что привело к приблизительно 2,45 миллионам смертей. Необходимы методы предотвращения или лечения инфекции SARS-CoV-2 и диагностические инструменты для диагностики инфекции SARS-CoV-2.
Краткое описание графических материалов
На фиг. 1A и 1B показано связывание антителами (1A) S303 (VH SEQ ID NO:63; VL SEQ ID NO:67) и (1B) S309 (VH SEQ ID NO:105; VL SEQ ID NO:168) по настоящему изобретению с рекомбинантным RBD (рецептор-связывающий домен) SARS-CoV-2, как описано в примере 2.
На фиг. 2A и 2B показана нейтрализация инфекции SARS-CoV-2 с помощью определенных антител по настоящему изобретению, как описано в примере 4.
На фиг. 3A-3I показана нейтрализация инфекции SARS-CoV-2, как описано в примере 4. На фиг. 3A показывает нейтрализацию с помощью донорской плазмы у выживших жертв SARS-CoV-1. На фиг. 3B-3D и 3I показана нейтрализация с помощью супернатанта из В-клеток, экспрессирующих определенные антитела по настоящему изобретению. На фиг. 3E-3H показана нейтрализация с помощью определенных рекомбинантных антител IgG1.
На фиг. 4A и 4B показано связывание супернатанта В-клеток, содержащего антитело, с белком SARS-CoV-2, экспрессируемым на клетках ExpiCHO (CHO - клетки яичника китайского хомячка), как описано в примере 1. Графики, показывающие профили связывания антител S300-S310, обозначены рамками.
На фиг. 5A и 5B показано связывание антител S311 и S312 в супернатанте культивированных В-клеток с SARS-CoV-1 и SARS-CoV-2, как описано в примере 6. Концентрации антител являются приблизительными. SARS S1 Sino: белок от Sino Biological. RBD2: RBD SARS-CoV-2, полученный собственными силами.
На фиг. 6A-6E показаны кривые (сверху) связывания некоторых антител по настоящему изобретению с RBD SARS-CoV-1 (SARS1) и RBD SARS-CoV-2 (SARS2), измеренные с помощью прибора Octet и кривые (снизу) значений KD. Значения KD для антител (например, менее 1,0×10-12 M) с очень сильным связыванием и медленной диссоциацией являются приблизительными. Эти данные и эксперименты описаны далее в Примере 3.
На фиг. 7 показана нейтрализация инфекции с помощью антител S304 (VH SEQ ID NO:79; VL SEQ ID NO:83) и S309 (VH SEQ ID NO:105; VL SEQ ID NO:168), по отдельности или в комбинации, против псевдотипированного SARS-CoV-2 вируса, как описано в примере 5.
На фиг. 8A-8K показаны кривые связывания некоторых антител с RBD SARS-CoV-1, RBD SARS-CoV-2 и эктодоменами различных коронавирусов, измеренных с помощью ELISA (иммуноферментный анализ). См. пример 8.
На фиг. 9 показана нейтрализация инфекции S309 rIgG1 (VH SEQ ID NO:105; VL SEQ ID NO:168) и S315 rIgG1 (VH SEQ ID NO:178; VL SEQ ID NO:182) против псевдотипированного SARS-CoV-2 вируса, как описано в примере 7.
На фиг. 10 показана нейтрализация инфекции с помощью полноразмерного S309 rIgG1 и S309 rFab (оба из которых содержат VH с SEQ ID NO:105 и VL с SEQ ID NO:168) против псевдотипированного SARS-CoV-2 вируса, как описано в примере 7.
На фиг. 11 показано связывание антитела S309 (VH SEQ ID NO:105; VL SEQ ID NO:168) со спайк-белком SARS-CoV-1 и SARS-CoV-2, экспрессируемым на клетках ExpiCHO, как описано в примере 9. Сложенные гистограммы графиков проточной цитометрии демонстрируют дозозависимое связывание антитела S309 с SARS-CoV и SARS-CoV-2.
На фиг. 12A и 12B показано зависимое от концентрации связывание, измеренное с помощью проточной цитометрии для определенных антител, как описано в примере 9. На фиг. 12A показано связывание с SARS-CoV-2. На фиг. 12B показано связывание с SARS-CoV-1.
На фиг. 13 показана нейтрализация инфекции с помощью антител S303 (VH SEQ ID NO:63; VL SEQ ID NO:67), S304 (VH SEQ ID NO:79; VL SEQ ID NO:83), S306 (VH SEQ ID NO:87; VL SEQ ID NO:91), S309 (VH SEQ ID NO:105; VL SEQ ID NO:168), S310 (VH SEQ ID NO:155; VL SEQ ID NO:159) и S315 (VH SEQ ID NO:178; VL SEQ ID NO:182) против псевдотипированного SARS-CoV-2 вируса, как описано в примере 4.
На фиг. 14A-14D показана аффинность/авидность связывания антител S309 (VH SEQ ID NO:105; VL SEQ ID NO:168), S303 (VH SEQ ID NO:63; VL SEQ ID NO:67), S304 (VH SEQ ID NO:79; VL SEQ ID NO:83) и S315 (VH SEQ ID NO:178; VL SEQ ID NO:182) с RBD SARS-CoV-1 (справа) и SARS-CoV-2 (слева), как описано в примере 10.
На фиг. 15А и 15В показана конкуренция пар антител по настоящему изобретению за связывание с RBD SARS-CoV-1 (фиг. 15А) и SARS-CoV-2 (фиг. 15В), как описано в примере 12. Для каждого графика ось x показывает время (от 0 до 1000 секунд), а ось y показывает связывание с RBD, измеренным с помощью BLI (биослойная интерферометрия) (от 0 до 3 нм). Первое антитело указано на левой стороне матрицы, а второе антитело - на верхней части матрицы. Пунктирные вертикальные линии на фиг. 15В иллюстрируют переключение от первого антитела ко второму антителу. Справа ("I" - "IV" на фиг. 15А, "II" и "IV" на фиг. 15В) находятся антигенные сайты, определенные с помощью структурной информации, анализа "ускользнувших" мутантов и связывания эпитопов на основе BLI.
На фиг. 16 показана способность S309 мешать связыванию RBD SARS-CoV-1 (слева) или SARS-CoV-2 (справа) с ACE2 (ангиотензинпревращающий фермент 2) человека (hACE2), как описано в примере 13. hACE2 загружали на сенсоры BLI с последующей инкубацией сенсоров с RBD отдельно или RBD в комбинации с антителом. Вертикальная пунктирная линия указывает на начало ассоциации RBD с антителом или без антитела. На графике слева антитело S230 использовали в качестве положительного контроля ингибирования связывания RBD SARS-CoV-1 с ACE2 на основании предыдущих исследований (см. Walls и др., Cell 176(5):1023-1039.e15 (2019)).
На фиг. 17А И 17В показаны антителозависимые эффекты некоторых антител по настоящему изобретению против модели инфицированных клеток, как описано в примере 14. На фиг. 17A показана антителозависимая цитотоксичность с использованием первичных NK-эффекторных клеток (NK - естественные киллеры) и экспрессирующих SARS-CoV-2- клеток ExpiCHO в качестве клеток-мишеней. Гистограмма справа показывает ADCC (антителозависимая клеточно-опосредованная цитотоксичность) для указанного антитела (антител), рассчитанного как площадь под кривой (AUC). На фиг. 17B показан антителозависимый клеточный фагоцитоз с использованием PBMC (мононуклеарные клетки периферической крови) в качестве фагоцитарных клеток и PKF67-меченых SARS-CoV-2-экспрессирующих ExpiCHO в качестве клеток-мишеней. Линейные графики показывают среднюю интенсивность флуоресценции (MFI) PBMC после инкубации с клетками-мишенями и антителами, определенную для одного репрезентативного донора с FcγRIIIa с высокой аффинностью (Fc гамма рецептор IIIa) (символы показывают среднее значение ±SD (стандартное отклонение) повторностей).
На фиг. 18A-18J показаны кривые связывания некоторых антител с RBD SARS-CoV-1, RBD SARS-CoV-2 и эктодоменами различных штаммов коронавирусов, измеренных с помощью ELISA. См. пример 8. Рекомбинантные mAb (моноклональное антитело) тестировали с помощью ELISA в диапазоне концентраций от 5 до 0,00028 мг/мл. RBD2: рецептор-связывающий домен SARS-CoV-2. RBD1: рецептор-связывающий домен SARS-CoV (также называемый в настоящем документе SARS-CoV-1). Спайк-белок: стабилизированный префузионный тример указанного коронавируса. Некоторые антитела рекомбинантно экспрессировались как IgG1 (иммуноглоулин IgG1) (rIgG1), а некоторые антитела рекомбинантно экспрессировались как IgG1 с мутацией MLNS (M428L и N434S (нумерация EU)) в Fc (rIgG1-LS).
На фиг. 19A и 19B показана способность некоторых антител препятствовать связыванию RBD с человеческим ACE2, как описано в примере 13. Человеческий ACE2 (hACE2) загружали на сенсоры BLI с последующей инкубацией сенсоров только с RBD или RBD в комбинации с рекомбинантным антителом. Вертикальная пунктирная линия указывает на начало загрузки RBD с антителом или без антитела. RBD: рецептор-связывающий домен. На фиг. 19A показано связывание RBD SARS-CoV-1 с ACE2. На фиг. 19В показано связывание RBD SARS-CoV-2 с ACE2.
На фиг. 20A и 20B показаны аффинность связывания и авидность антитела S309 IgG (фиг. 20A) по сравнению с S309 Fab (фиг. 20B) для RBD SARS-CoV-1 (внизу каждой фиг.) и RBD SARS-CoV-2 (вверху каждой фиг.), как описано в примере 10. Как для IgG, так и для Fab: VH SEQ ID NO:105; VL SEQ ID NO:168. RBD загружали на цилиндры BLI и измеряли ассоциацию различных концентраций S309-IgG-MLNS или S309 Fab. Вертикальные пунктирные линии указывают на начало фазы диссоциации, когда цилиндры BLI были переключены на буфер.
На фиг. 21A-21C показана реактивность антител S304 (VH SEQ ID NO:79; VL SEQ ID NO:83), S306 (VH SEQ ID NO:87; VL SEQ ID NO:91), S309 (VH SEQ ID NO:105; VL SEQ ID NO:168) и S310 (VH SEQ ID NO:155; VL SEQ ID NO:159) против SARS-CoV-2, как описано в примере 15. На фиг. 21A показана реактивность антител S304, S306, S309 и S310 против лизата, экстрагированного T×100, клеток Vero E6, инфицированных SARS-CoV-2. На фиг. 21B показана реактивность тех же антител против лизата, экстрагированного SDS, клеток Vero E6, инфицированных SARS-CoV-2. На фиг. 21C показаны сыворотки выздоравливающего человека от SARS-CoV-1 против экстрагированного T×100 или экстрагированного SDS лизата клеток Vero E6, инфицированных SARS-CoV-2. На фиг. 21A и 21B также показаны данные для сравнительного антитела LCA57, которое является специфичным для спайк-белка MERS-CoV (Corti и др. PNAS 112(33):10473-10478 (2015).
На фиг. 22A-22D показана нейтрализация инфекции с помощью SARS-CoV-2 антител, что оценивалось по ингибированию экспрессии нуклеопротеина (NP) через 24 и 45 часов после инфицирования. См. пример 16. На фиг. 22A показана нейтрализация инфекции SARS-CoV-2 с помощью S304 S304 (VH SEQ ID NO:79; VL SEQ ID NO:83). На фиг. 22B показана нейтрализация инфекции SARS-CoV-2 с помощью S309 (VH SEQ ID NO:105; VL SEQ ID NO:168. На фиг. 22C показана нейтрализация инфекции SARS-CoV-2 комбинацией S304 и S309. На фиг. 22D показана контрольная нейтрализация инфекции SARS-CoV-2 с помощью сравнительного антитела LCA57, которое является специфичным для спайк-белка MERS-CoV (коронавирус ближневосточного респираторного синдрома (Corti и др. PNAS 112(33):10473-10478 (2015).
На фиг. 23 показана нейтрализация инфекции с помощью антител S309 (VH SEQ ID NO:105; VL SEQ ID NO:168) и S315 (VH SEQ ID NO:178; VL SEQ ID NO:182) отдельно или в комбинации против псевдотипированного SARS-CoV-2 вируса, как описано в примере 5.
На фиг. 24А И 24В показаны антителозависимые эффекты некоторых антител по настоящему изобретению против модели инфицированных клеток, как описано в примере 14. На фиг. 24A показана антителозависимая клеточно-опосредованная цитотоксичность (ADCC) с использованием первичных NK-эффекторных клеток и экспрессирующих SARS-CoV-2- клеток ExpiCHO в качестве клеток-мишеней. График показывает процент уничтожения клеток-мишеней после инкубации с антителом или комбинацией антител, показанных в условных обозначениях. На фиг. 24В показана ADCC для указанного антитела (антител), рассчитанная как площадь под кривой (AUC). Слева: AUC, определенная с использованием клеток с генотипом FcγRIIIa VV; справа: AUC, определенная для клеток с генотипом FcγRIIIa FF или FV.
На фиг. 25А и 25В показаны дополнительные антителозависимые эффекты некоторых антител по настоящему изобретению, как описано в примере 14. На фиг. 25A показан антителозависимый клеточный фагоцитоз (ADCP) с использованием PBMC в качестве фагоцитарных клеток и PKF67-меченых SARS-CoV-2-экспрессирующих ExpiCHO клетки в качестве клеток-мишеней. Графики показывают среднюю интенсивность флуоресценции (MFI) PBMC после инкубации с клетками-мишенями и антителами, определенную для одного репрезентативного донора c FcγRIIIa с высокой аффинностью (символы показывают среднее значение ±SD повторностей). На фиг. 24В показана ADCC для указанного антитела (антител), рассчитанная как площадь под кривой (AUC).
На фиг. 26 показано связывание антитела, измеренное с помощью проточной цитометрии. Связывание антитела S309 со спайк-белком SARS-CoV-2, экспрессируемым в клетках Expi-CHO, обнаруживали с помощью сортировки клеток с активированной флуоресценцией.
На фиг. 27 показано связывание антитела S309 (помеченного как "11" в ключе фигуры) и четырех сконструированных вариантов S309 (помеченного как "12" - "15", соответственно) с S-белком, измеренное с помощью проточной цитометрии. См. пример 9. Четыре сконструированных варианта антител являются следующими: S309 N55Q содержит мутацию N55Q в CDRH2, что приводит к последовательности варианта VH (SEQ ID NO:113), и последовательность VL дикого типа (SEQ ID NO:168) S309; S309 W50F содержит последовательность VH варианта W50F (SEQ ID NO: 129) и последовательность VL дикого типа (SEQ ID NO:168) S309; S309 W105F содержит последовательность VH варианта W105F (SEQ ID NO: 119) и последовательность VL дикого типа (SEQ ID NO:168) S309; и S309 W50F/G56A/W105F содержит последовательность варианта VH W50F/G56A/W105F (SEQ ID NO:172) и последовательность VL дикого типа S309. На фиг. 27 S309 N55Q помечен как «12», S309 W50F помечен как «13», S309 W105F помечен как «14», а S309 W50F-G56A-W105F помечен как «15». Связывание антитела со спайк-белком SARS-CoV-2, экспрессируемым на клетках Expi-CHO, обнаруживали с помощью флуоресцентно меченого вторичного антитела. Показаны данные двух экспериментов.
На фиг. 28 показана нейтрализация инфекции с помощью антитела S309 (называемое в фиг. "Вариант-11 (wt (дикий тип))") и четырех вариантных антител S309 против псевдотипированных SARS-CoV-2 вирусов, как описано в примере 19. На фиг. 28S309 N55Q помечен как «Вариант-12», S309 W50F помечен как «Вариант-13», S309 W105F помечен как «Вариант-14», а S309 W50F-G56A-W105F помечен как «Вариант-15». Псевдотипированные вирусы представляют собой псевдотипированные VSV (вирус везикулярного стоматита) со спайк-белком SARS-CoV-2.
На фиг. 29 показан обзор результатов анализов связывания и нейтрализации псевдовирусов с помощью антитела S309 ("S309-WT") и четырех сконструированных вариантов S309 ("N55Q"; "W50F"; "W105F"; "W50F/G56A/W105F"). Пунктирная горизонтальная линия показывает изменение функции сконструированного варианта по сравнению с базовой линией S309-WT. Заштрихованные различными способами полосы демонстрируют связывание с гликозилированным RBD, измеренное с помощью SPR (поверхностный плазмонный резонанс), связывание с дегликозилированным RBD, измеренное с помощью SPR, связывание с антигенэкспрессирующими клетками, измеренное с помощью FACS (cортировка клеток с активированной флуоресценцией), и нейтрализацию, измеренную с помощью псевдовирусов SARS-CoV-2.
На фиг. 30A-30F показана кинетика связывания примерных антител с гликозилированным или дегликозилированным RBD SARS-CoV-2, измеренная с помощью SPR. См. пример 18. Были протестированы антитела S309 (имеющие аминокислотные последовательности VH (SEQ ID NO:105) и VL (SEQ ID NO: 168) S309 дикого типа), S309 N55Q, S309 W50F, S309 W105F и S309 W50F/G56A/W105F. На фиг. 30A показана кинетика связывания антитела S309 дикого типа (2 повторных эксперимента). На фиг. 30B показана кинетика связывания S309 N55Q (снизу) по сравнению с антителом S309 дикого типа (сверху). На фиг. 30C показана кинетика связывания S309 W50F (снизу) по сравнению с антителом S309 дикого типа (сверху). На фиг. 30D показана кинетика связывания S309 W105F (снизу) по сравнению с антителом S309 дикого типа (сверху). На фиг. 30E показано связывание S309 W50F/G56A/W105F (снизу) по сравнению с антителом дикого типа S309 (сверху). На фиг. 30F показано связывание S309 W50F/G56A/W105F с использованием 10-минутного периода инъекции (сверху) или 3-минутного периода инъекции (снизу).
На фиг. 31 показана активация FcγRIIIa с высокой аффинностью (158V) (слева) или FcγRIIIa с низкой аффинностью (158F) (справа) антителами S303 (VH SEQ ID NO:63; VL SEQ ID NO:67), S304 (VH SEQ ID NO:79; VL SEQ ID NO:83), S306 (VH SEQ ID NO:87; VL SEQ ID NO:91), S309 (VH SEQ ID NO:105; VL SEQ ID NO:168) и комбинацией S309 и S315, наряду со сравнительным антителом S230. См. пример 20. Активацию измеряли с использованием S-экспрессирующих SARS-CoV-2 клеток ExpiCHO в качестве клеток-мишеней и репортерных клеток Jurkat, стабильно трансфицированных репортерным геном люциферазы, управляемым NFAT (ядерный фактор активированных Т-клеток). Активация FcγRIIIa приводит к NFAT-опосредованной экспрессии репортерного гена люциферазы. Результаты получены в результате одного эксперимента, одного или двух измерений на mAb (моноклональное антитело).
На фиг. 32 показана активация FcγRIIa с помощью примерных антител S303 (VH SEQ ID NO:63; VL SEQ ID NO:67), S304 (VH SEQ ID NO:79; VL SEQ ID NO:83), S306 (VH SEQ ID NO:87; VL SEQ ID NO:91), S309 (VH SEQ ID NO:105; VL SEQ ID NO:168) и комбинации S309 и S315, наряду со сравнительным моноклональным антителом S230. См. пример 20. Активацию измеряли с использованием S-экспрессирующих SARS-CoV-2 клеток ExpiCHO в качестве клеток-мишеней и репортерных клеток Jurkat, стабильно трансфицированных репортерным геном люциферазы, управляемым NFAT (ядерный фактор активированных Т-клеток). Активация FcγRIIa приводит к NFAT-опосредованной экспрессии репортерного гена люциферазы.
На фиг. 33A и 33B показано связывание антител S303 (VH SEQ ID NO:63; VL SEQ ID NO:67), S304 (VH SEQ ID NO:79; VL SEQ ID NO:83), S306 (VH SEQ ID NO:87; VL SEQ ID NO:91), S309 (VH SEQ ID NO:105; VL SEQ ID NO:168), S310 (VH SEQ ID NO:155; VL SEQ ID NO:159) и S315 (VH SEQ ID NO:178; VL SEQ ID NO:182), наряду со сравнительными антителами S110, S230 и S109, с S-белком, экспрессируемым на поверхности клетки. См. пример 9. На фиг. 33A показано связывание с клетками ExpiCHO, трансфицированными S-белком SARS-CoV-2. На фиг. 33В показано связывание с клетками ExpiCHO, трансфицированными S-белком SARS-CoV-1. Среднюю интенсивность флуоресценции измеряли с помощью проточной цитометрии для каждого антитела. Исследованные концентрации антител указаны по оси X.
На фиг. 34 показана нейтрализация инфекции с помощью примерных антител S303 (VH SEQ ID NO:63; VL SEQ ID NO:67), S304 (VH SEQ ID NO:79; VL SEQ ID NO:83), S306 (VH SEQ ID NO:87; VL SEQ ID NO:91), S309 (VH SEQ ID NO:105; VL SEQ ID NO:168), S310 (VH SEQ ID NO:155; VL SEQ ID NO:159) и S315 (VH SEQ ID NO:178; VL SEQ ID NO:182) против псевдотипированного SARS-CoV-2 вируса, как описано в примере 4. См. пример 4.
На фиг. 35A и 35B показана консервация остатков S-белка, как описано в примере 21. На фиг. 35A показаны варианты спайк-белка, встречающиеся с частотой n более 1 в виде сфер, отображаемых в закрытой (слева) и открытой (справа) форме полного тримерного спайк-эктодомена. RBD и другие домены спайк-белка показаны как указано. Показано 40 мутаций (из 2229 в общей сложности). Только остаток 367 (представляет собой 8) выделен в RBD, но не остатки 476 (n представляет собой 7) и 483 (n представляет собой 17). На фиг. 35B показано преобладание вариантов спайк-гликопротеинов по аминокислотам. Каждая точка является отдельным вариантом. Показаны местоположения домена A и RBD. Варианты, соответствующие пороговому значению частоты 0,1%, являются такими, как указано.
На фиг. 36 показана нейтрализация SARS-CoV-2-MLV (MLV - вирус лейкоза мышей) с помощью антитела S309 (VH SEQ ID NO:105; VL SEQ ID NO:168) в комбинации с эквивалентным количеством антитела S304 (VH SEQ ID NO:79; VL SEQ ID NO:83). Для коктейлей из антител концентрация, показанная на оси x, равна концентрации отдельных антител. См. пример 4.
На фиг. 37 показана нейтрализация SARS-CoV2-MLV с помощью антитела S309 (VH SEQ ID NO:105; VL SEQ ID NO:168) в сочетании с эквимолярным количеством антител S315 (VH SEQ ID NO:178; VL SEQ ID NO:182). Для коктейлей из антител концентрация, показанная на оси x, равна концентрации отдельных антител. См. пример 4.
На фиг. 38A-38D показано связывание определенных антител с RBD SARS-CoV-2 и SARS-CoV-1, как описано в примере 6. Антитела экспрессировали рекомбинантно и связывание анализировали с использованием ELISA. 96-луночные планшеты для ELISA покрывали RBD SARS-CoV-2 (полученный собственными силами; остатки 331-550 спайк-белка из BetaCoV/Wuhan-Hu-1/2019, кат. № MN908947) при 10 мкг/мл и RBD SARS-CoV-1 (Sino Biological, 40150-V08B1) при 1 мкг/мл. После блокирования 1% BSA (бычий сывороточный альбумин) в PBS (фосфатно-солевой буфер) антитела добавляли в планшеты и инкубировали при комнатной температуре в течение одного часа. Планшеты промывали и добавляли вторичные антитела козы против IgG-AP человека (Southern Biotechnology, 2040-04). Субстрат п-нитрофинилфосфат (pNPP, Sigma-Aldrich, 337 71768) использовали для создания цвета. OD405 анализировали на планшетном ридере ELx808IU (Biotek). Слева на каждой фигуры показано связывание с RBD SARS-CoV-2, а справа показано связывание с RBD SARS-CoV-1.
На фиг. 39А и 39В показано связывание определенных антител со спайк-белком SARS-CoV-2 и SARS-CoV-1, как описано в примере 9. Клетки Expi-CHO временно трансфицировали phCMV1-SARS-CoV-2-S, SARS-спайк-pcDNA (ДНК (дезоксирибонуклеиновая кислота) елонирующая плазмиду).3 (штамм SARS) или пустой phCMV1 с использованием Expifectamine CHO Enhancer. Через два дня после трансфекции клетки собирали для иммуноокрашивания антителами. Для обнаружения использовали меченный Alexa647 вторичное антитело против Fc IgG человека. Связывание антител с трансфицированными клетками анализировали с помощью проточной цитометрии с использованием клеточного анализатора ZE5 (BioRad). На фиг. 39A показано связывание рекомбинантного антитела S300 (VH: SEQ ID NO:1; VL: SEQ ID NO:5). На фиг. 39B показано связывание рекомбинантного антитела S307. Символы показывают значения одного измерения. Слева на каждой фигуре показаны данные, представленные как % положительных клеток, а на правой панели показаны данные, представленные как средняя интенсивность флуоресценции (MFI).
На фиг. 40A и 40B показано связывание типовых антител с S-гликопротеинами SARS-CoV-2 (фиг. 40A) или SARS-CoV-1 (фиг. 40B), экспрессируемыми на поверхности клеток ExpiCHO, как описано в примере 9. Символы представляют собой повторности одного эксперимента.
На фиг. 41A и 41B показаны аффинность связывания и авидность IgG S309 (фиг. 41A) и Fab S309 (фиг. 41B) по отношению к RBD SARS-CoV-2 (сверху) или спайк-белку SARS-CoV-2 (снизу). Как для IgG, так и для Fab: VH SEQ ID NO:105; VL SEQ ID NO:168. См. Пример 11. Биотинилированный RBD SARS-CoV-2 или биотинилированный префузионный тример S-эктодомена SARS-CoV-2 загружали в биосенсоры стрептавидина и измеряли ассоциацию различных концентраций S309-IgG-MLNS (содержащего мутации M428L и N434S Fc (нумерация EU)) или Fab S309. Вертикальные пунктирные линии указывают на начало фазы диссоциации, когда биосенсоры были переключены на буфер.
На фиг. 42А И 42В показано связывание антител S303, S304, S306, S309, S310 и S315, наряду со сравнительными антителами S110, S124, S230 и S109, с S-белком, экспрессируемым на поверхности клетки. См. пример 9. На фиг.42A показано связывание с клетками ExpiCHO, трансфицированными S-белком SARS-CoV-2. На фиг. 42В показано связывание с клетками ExpiCHO, трансфицированными S-белком SARS-CoV-1. Среднюю интенсивность флуоресценции измеряли с помощью проточной цитометрии для каждого антитела. Исследованные концентрации антител указаны по оси X.
На фиг. 43 показано сохранение остатков спайк-белка, как описано в примере 21. Варианты спайк-белка, подтверждаемые по меньшей мере двумя последовательностями, как указано, сферами, отображенными в закрытой (слева) и открытой (справа) форме полного тримерного спайк-эктодомена. RBD и другие домены спайк-белка показаны как указано. Показаны 171 вариант (из 11 839 проанализированных последовательностей спайк-белка).
На фиг. 44A-44C показан отбор на устойчивость к SARS-CoV-2, как описано в примере 23. На фиг. 44А показана блок-схема, иллюстрирующая способ отбора на устойчивость. На фиг. 44B показана временная шкала, иллюстрирующая процедуру, используемую для каждого пассажа процесса отбора на устойчивость. На фиг. 44C показаны результаты отбора на устойчивость SARS-CoV-2 к антителу S309 N55Q MLNS GAALIE, содержащего VH согласно SEQ ID NO:113 и VL согласно SEQ ID NO:168, с мутациями G236A, A330L, I332E, M428L и N434S в Fc (нумерация EU).
На фиг. 45 показана антителозависимая цитотоксичность некоторых антител по настоящему изобретению с использованием первичных NK-клеток в качестве эффекторных клеток и SARS-CoV-2-экспрессирующих клеток ExpiCHO в качестве клеток-мишеней. См. пример 14. На графике показан процент уничтожения клеток-мишеней после инкубации с антителами S309 LS (также называемое в настоящем документе S309 MLNS), S309 GRLR (G236R/L328R; вариант, не связывающий FcR) или S309 LS GAALIE (также называемое в настоящем документе S309 MLNS GAALIE, содержащем мутации Fc G236A, A330L, I332E, M428L и N434S (нумерация EU)).
На фиг. 46 показана нейтрализация инфекции SARS-CoV-2 в клетках легких человека Calu-3 (клеточная линия рака легкого человека) и клетках VeroE6 с помощью антитела S309 N55Q MLNS, как описано в примере 24.
На фиг. 47 показана нейтрализация инфекции SARS-CoV-2 с помощью антитела S309 (VH SEQ ID NO:105; VL SEQ ID NO:168), как обнаружено с помощью нанолюциферазного анализа. См. пример 25. Ось X показывает концентрацию антител. Три кривые представляют собой анализы с использованием трех различных концентраций вируса в единицах MOI (множественность инфекции), как показано на рисунке справа. Данные собирали через шесть часов после инфицирования вирусом SARS-CoV-2. Рассчитанные значения IC50 (концентрация полумаксимального ингибирования) для каждого MOI показаны в полях под графиком.
На фиг. 48A и 48B показана нейтрализация инфекции SARS-CoV-2 с помощью антитела S309 (VH SEQ ID NO:105; VL SEQ ID NO:168), как проанализировано с помощью IFA (иммунофлюоресцентный анализ). См. пример 25. Данные собирали через шесть часов после инфицирования вирусом SARS-CoV-2. На фиг. 48А показаны фотографии репрезентативных лунок, на которых ядра клеток окрашивали синим цветом, а нуклеокапсиды SARS-CoV-2 окрашивали красным цветом, что более ярко проявляется на фотографиях. Каждое красное пятно на каждом изображении представляет собой отдельную инфицированную клетку. Нейтрализация инфекции с помощью антитела S309 может наблюдаться как уменьшение количества ярких пятен, указывающих на инфицированные клетки, так как концентрация S309 была увеличена. Концентрации антител показаны сверху, а концентрации вирусов в единицах MOI (множественность инфекции) показаны слева. На фиг. 48B показаны количественные данные из анализа IFA. Рассчитанные значения IC50 для каждого MOI показаны в полях под графиком.
На фиг. 49 показана нейтрализация инфекции SARS-CoV-2 с помощью антител S309 N55Q LS (также называемое в настоящем документе S309 N55Q MLNS, содержащего мутации Fc M428L/N434S (нумерация EU)) и S309 N55Q LS GAALIE (также называемое в настоящем документе S309 N55Q MLNS GAALIE, содержащего мутации Fc G236A, A330L, I332E, M428L и N434S (нумерация ЕС)). См. пример 26. Каждый из S309 N55Q LS и S309 N55Q LS GAALIE содержит VH, имеющий аминокислотную последовательность, представленную в SEQ ID NO:113, и VL, имеющий аминокислотную последовательность, представленную в SEQ ID NO:168. Данные представляют собой среднее значение четырех повторностей, +/- стандартное отклонение. Показанный график является репрезентативным для трех независимых экспериментов.
На фиг. 50A и 50B показана нейтрализация инфекции псевдотипированными SARS-CoV-2 вирусами с использованием антител S309 N55Q LS (также называемое в настоящем документе S309 N55Q MLNS, содержащего мутации Fc M428L/N434S (нумерация EU)) (фиг. 50A) и S309 N55Q LS GAALIE (также называемое в настоящем документе S309 N55Q MLNS GAALIE, содержащего мутации Fc G236A, A330L, I332E, M428L и N434S (нумерация EU)) (фиг. 50B). См. пример 27. Псевдотипированные вирусы представляют собой псевдотипированные VSV со спайк-белком SARS-CoV-2. Данные представляют собой среднее значение четырех повторностей, +/- стандартное отклонение. Каждый показанный график является репрезентативным для четырех независимых экспериментов.
На фиг. 51A и 51B показано связывание антител S309 N55Q MLNS (фиг. 51A) и S309 N55Q MLNS GAALIE (фиг. 51B) с RBD SARS-CoV-2, измеренное с помощью поверхностного плазмонного резонанса (SPR). См. пример 28. Значения взяты из двух независимых экспериментов.
На фиг. 52A и 52B показано связывание антител S309 N55Q LS (также называемое в настоящем документе S309 N55Q MLNS, содержащего мутации Fc M428L/N434S (нумерация EU)) (фиг. 52A) и S309 N55Q LS GAALIE (также называемое в настоящем документе S309 N55Q MLNS GAALIE, содержащего мутации Fc G236A, A330L, I332E, M428L и N434S (нумерация EU)) (фиг. 52B) с экспрессируемым на поверхности клетки спайк-белком SARS-CoV-2, как измерено с помощью проточной цитометрии. См. пример 29. Данные выражены в виде процента клеток, идентифицированных как положительные на связывание антител. Представленные результаты получены в ходе одного эксперимента и являются репрезентативными для трех независимых отдельных проведенных экспериментов.
На фиг. 53 показано связывание антител S309 N55Q MLNS и S309 N55Q MLNS GAALIE с FcγRIIa человека (как аллеля R131 с низкой аффинностью, так и H131 с высокой аффинностью), FcγRIIIa (как аллеля F158 с низкой аффинностью, так и V158 с высокой аффинностью) и FCγRIIb с использованием SPR. См. пример 30. Биотинилированные очищенные FcγR захватывали на поверхности сенсорного чипа перед инъекцией антитела. Профили ассоциации и диссоциации (разделенные вертикальной пунктирной линией на каждом графике) измеряли в режиме реального времени как изменение сигнала SPR.
На фиг. 54 показано связывание антител S309 (VH: SEQ ID NO:105; VL: SEQ ID NO:168) LS (также называемое в настоящем документе S309 MLNS, содержащего мутации Fc M428L/N434S), S309 N55Q (VH: SEQ ID NO:113; VL: SEQ ID NO:168) LS (также называемое в настоящем документе S309 N55Q MLNS, содержащего мутации Fc M428L/N434S (нумерация EU)) и S309 N55Q LS GAALIE (также называемое в настоящем документе S309 N55Q MLNS GAALIE, содержащего мутации Fc G236A, A330L, I332E, M428L и N434S (нумерация EU)) с компонентом комплемента C1q, что измеряется с помощью BLI на приборе Octet. См. пример 31. Профили ассоциации и диссоциации (разделенные вертикальной пунктирной линией на графике) измеряли в режиме реального времени как изменение интерференционной картины.
На фиг. 55A-55D показана активация in vitro FcγR человека антителами S309 (VH: SEQ ID NO:105; VL: SEQ ID NO:168) LS (также называемое в настоящем документе S309 MLNS, содержащим мутации Fc M428L/N434S), S309 N55Q (VH: SEQ ID NO:113; VL: SEQ ID NO:168) LS (также называемое в настоящем документе как S309 N55Q MLNS, содержащим мутации Fc M428L/N434S) (нумерация EU) и S309 N55Q LS GAALIE (также называемое настоящем документе S309 N55Q MLNS GAALIE, содержащим мутации Fc G236A, A330L, I332E, M428L и N434S (нумерация EU), наряду с отрицательным контролем антител S309 GRLR. См. пример 32. Клетки CHO, стабильно трансфицированные спайк-белком SARS-CoV-2, служили в качестве мишеней антител. Серийные разведения антитела инкубировали с клетками-мишенями при комнатной температуре в течение 15 минут. Эффекторные клетки Jurkat, экспрессирующие указанный FcγR и сконструированные с помощью NFAT-опосредованного репортера люциферазы, ресуспендировали в буфере для анализа, а затем добавляли в планшеты для анализа. После инкубации при 37 °С в течение 18 часов добавляли реагент для люциферазного анализа Bio-Glo™ (Promega) и количественно определяли люминесценцию с помощью лиминометра (Bio-Tek). На графиках показана активация FcγRIIa человека (сверху слева), FcγRIIb (сверху справа), FcγRIIIa аллеля F158 с низкой аффинностью (снизу слева) и FCγRIIb аллеля V158 с высокой аффинностью (снизу справа). Показаны средние значения +/- стандартного отклонения повторностей.
На фиг. 56A и 56B показано опосредованное NK-клетками уничтожение (ADCC) клеток, экспрессирующих спайк-белок SARS-CoV-2, in vitro в присутствии антител S309 (VH: SEQ ID NO:105; VL: SEQ ID NO:168) LS (также называемое в настоящем документе S309 MLNS, содержащего мутации Fc M428L/N434S ), S309 N55Q (VH: SEQ ID NO:105; VL: SEQ ID NO:168) LS (также называемое в настоящем документе S309 N55Q MLNS, содержащего мутации Fc M428L/N434S (нумерация EU)) или S309 N55Q LS GAALIE (также называемое в настоящем документе как S309 N55Q MLNS GAALIE, содержащего Fc мутации G236A, A330L, I332E, M428L и N434S (нумерация EU)) или контрольного антитела S309 GRLR. См. пример 33. Серийные разведения антитела (серийно разбавленные в 10 раз в среде AIM-V от 40 000 нг/мл до 0,075 нг/мл) инкубировали с клетками CHO-CoV-2 со спайк-белком в течение 10 минут перед смешиванием с NK-клетками в течение 4 часов. NK-клетки свежевыделены от двух доноров, ранее генотипированных для гомозиготной экспрессии низкой аффинности (F/F158; фиг. 56A) или высокой аффинности (V/V158; фиг. 56B). ADCC измеряли с использованием анализа высвобождения LDH (лактатдегидрогеназа). Показаны средние значения +/- стандартное отклонение четырех повторностей.
На фиг. 57 показан моноцит-опосредованный фагоцитоз (ADCC) клеток, экспрессирующих спайк-белок SARS-CoV-2, in vitro в присутствии антител S309 (VH: SEQ ID NO:105; VL: SEQ ID NO:168) LS (также называемое в настоящем документе S309 MLNS, содержащего мутации Fc M428L/N434S), S309 N55Q (VH: SEQ ID NO:113; VL: SEQ ID NO:168) LS (также называемое в настоящем документе S309 N55Q MLNS, содержащего мутации Fc M428L и N434S (нумерация EU)) или S309 N55Q LS GAALIE (также называемое в настоящем документе как S309 N55Q MLNS GAALIE, содержащего Fc мутации G236A, A330L, I332E, M428L и N434S (нумерация EU)) или контрольного антитела S309 GRLR. См. пример 33. Антитела инкубировали с PKH67-мечеными клетками со спайк-белком CHO-CoV-2-в течение 10 минут перед смешиванием со свежевыделенными клетками PBMC, меченными Cell Trace Violet. Активность ADCP измеряли после инкубации в течение ночи с помощью проточной цитометрии в процентах от CD14+ моноцитов, которые были двойными положительными в отношении PKH67 и cell trace violet. Показаны средние значения +/- стандартного отклонения повторностей.
На фиг. 58A и 58B показано, что антитело S309 (VH: SEQ ID NO:105; VL: SEQ ID NO:168) ингибирует слияние клеток, опосредованное спайк-белком SARS-CoV-2. См. пример 34. На фиг. 58A показаны микрофотографии клеток, сконструированных для сверхэкспрессии спайк-белка SARS-CoV-2 в присутствии (снизу) или в отсутствии (сверху) S309. На фиг. 58B показаны количественные данные из анализа ингибирования слияния при различных концентрациях антитела.
На фиг. 59 представлены данные анализа на бляшкообразующие единицы; (FFU), демонстрирующие, что вариант антител S309 N55Q не вызывают опосредованного антителами усиления репликации SARS-CoV-2 в PBMC, полученных от доноров, или дендритных клетках человека. См. пример 35.
На фиг. 60 показана экспрессия (иммунофлуоресценция) трансгенов DC-SIGN (молекулы межклеточной адгезии дендритных клеток 3-захватывающего неинтегрина)/L-SIGN (молекула межклеточной адгезии клеток печени/лимфатического узла 3-захватывающего неинтегрина), DC-SIGN и ACE2 в клетках HEK293T (эмбриональная почка человека 293, экспрессирующая большой Т-антиген SV40), сконструированных для сверхэкспрессии указанного белка. См. пример 37.
На фиг. 61 показаны уровни инфекции псевдовирусом VSV в клетках HEK293T дикого типа и в клетках HEK293T, сконструированных для сверхэкспрессии DC-SIGN, L-SIGN или ACE2. Псевдовирус экспрессировал рекомбинантный спайк-белок SARS-CoV-2 с репортером люциферазы. См. пример 37.
На фиг. 62 показана нейтрализация моноклональным антителом S309 (VH SEQ ID NO:105, VL SEQ ID NO:168) инфекции псевдовирусом VSV в клетках HEK293T, сконструированных для сверхэкспрессии DC-SIGN, L-SIGN или ACE2. В этом примере антитело S309 содержит мутации Fc M428L и N434S (нумерация EU). См. пример 37.
На фиг. 63 показаны уровни инфекции живым SARS-CoV-2 в клетках HEK293T дикого типа и в клетках HEK293T, сконструированных для сверхэкспрессии DC-SIGN, L-SIGN или ACE2. Инфекцию определяли с использованием рекомбинантного S-белка с репортером люциферазы. См. пример 37.
На фиг. 64 показана нейтрализация примерным моноклональным антителом S309 (VH SEQ ID NO:105, VL SEQ ID NO:168) инфекции живым SARS-CoV-2 в клетках HEK293T, сконструированных для сверхэкспрессии DC-SIGN, L-SIGN или ACE2. В этом примере антитело S309 содержит мутации Fc M428L и N434S (нумерация EU). См. пример 37.
На фиг. 65показана экспрессия (иммунофлуоресценция) трансгенов L-SIGN, DC-SIGN, SIGLEC1 и ACE2 в клетках HEK293T, сконструированных для сверхэкспрессии указанного белка (белков). См. пример 37.
На фиг. 66 показаны уровни инфекции живым SARS-CoV-2 в клетках HEK293T дикого типа и в клетках HEK293T, сконструированных для сверхэкспрессии DC-SIGN, L-SIGN, SIGLEC-1 или ACE2. Инфекцию определяли с использованием рекомбинантного S-белка с репортером люциферазы. См. пример 37.
На фиг. 67 показана нейтрализация примерным моноклональным антителом S309 (VH SEQ ID NO:105, VL SEQ ID NO:168) инфекции живым вирусом SARS-CoV-2 в клетках HEK293T, сконструированных для сверхэкспрессии DC-SIGN, L-SIGN, SIGLEC-1 или ACE2. В этом примере антитело S309 содержит мутации Fc M428L и N434S (нумерация EU). См. пример 37.
На фиг. 68A и 68B показан анализ экспрессии рецепторных белков, включая белки CD209 (DC-SIGN) и SIGLEC в нескольких типах клеток. Размер точки коррелирует с процентом клеток указанного типа, которые экспрессируют белок, а интенсивность затенения точки коррелирует с уровнем экспрессии белка. См. пример 37.
На фиг. 69 показана инфекция, вызванная живым SARS-CoV-2, экспрессирующим N-люциферазу в клетках HEK293T («исходные») или клетках HEK293T, стабильно экспрессирующих DC-SIGN, L-SIGN, SIGLEC-1 или ACE2. Данные представляют собой эксперименты по тестированию SARS-CoV-2 при трех множественностях инфекций (MOI). См. пример 37.
На фиг. 70 показана инфекция VSV псевдотипированным SARS-CoV-2 в клетках HEK293T, клетках HeLa и клетках MRC5, временно трансдуцированных лентивирусом для экспрессии DC-SIGN, L-SIGN, SIGLEC-1 или ACE2. Неинфицированные клетки показаны как отрицательный контроль. См. пример 37.
На фиг. 71A и 71B показано связывание (измеренное с помощью биослойной интерферометрии) S309, S309 N55Q MLNS, S309 N55Q MLNS GAALIE (фиг. 71A) и сравнительных антител REGN10933 и REGN10987 (фиг. 71B) дикого типа с мутировавшими вариантами RBD. См. пример 39.
На фиг. 72 показана нейтрализация SARS-CoV-2 ("WT" представляет собой Wuhan-Hu-1; "UK" представляет собой SARS-CoV-2 вариант B.1.1.7; и "SA" представляет собой вариант B.1.351) псевдовируса MLV в клетках Vero-E6 с помощью антител S309, как описано в примере 39. Также оценивали сравнительные антитела REGN10987, REGN10933 и комбинацию REGN10987 + REGN10933.
На фиг. 73A-73D показано, что S309 (VH SEQ ID NO:105; VL SEQ ID NO:168) обеспечивает надежную защиту против инфекции in vivo SARS-CoV-2. Сирийским хомякам вводили указанное количество mAb за 48 часов до интраназальной инфекции SARS-CoV-2. (A) Количественное определение вирусной РНК (рибонуклеиновая кислота) в легких через 4 дня после инфицирования. (B) Количественное определение реплицирующего вируса в гомогенатах легких, собранных через 4 дня после инфицирования с использованием анализа TCID50 (доза вируса, вызывающая цитопатический эффект у 50 % зараженных культур клеток). (C) Гистологический показатель легочной ткани оценивали через 4 дня после инфицирования. (D) Концентрация mAb, измеренная в сыворотке до инфицирования (день 0), обратно коррелирует с вирусной нагрузкой РНК в легком через 4 дня после инфицирования. См. пример 38.
Подробное описание изобретения
В настоящем документе предложены антитела и антигенсвязывающие фрагменты, которые способны связываться с SARS-CoV-2 (например, с поверхностным гликопротеином SARS-CoV-2 и/или RBD, как описано в настоящем документе, в вирионе SARS-CoV-2 и/или экспрессируемыми на поверхности клетки-хозяина, такой как клетка, инфицированная SARS-CoV-2). Клетка-хозяин может представлять собой, например, клетку легкого, клетку СНО (такую как, например, клетка ExpiCHO, трансфицированная для экспрессии поверхностного гликопротеина) или т. п. В некоторых вариантах осуществления раскрытые в настоящем документе антитела и антигенсвязывающие фрагменты могут нейтрализовать инфекцию SARS-CoV-2 на модели инфекции in vitro и/или у субъекта-человека. Также предложены полинуклеотиды, кодирующие антитела и антигенсвязывающие фрагменты, векторы, клетки-хозяева и связанные композиции, а также способы применения антител, нуклеиновых кислот, векторов, клеток-хозяев и связанных композиций для лечения (например, уменьшения, задержки, устранения или предотвращения) инфекции SARS-CoV-2 у субъекта и/или для получения лекарственного средства для лечения инфекции SARS-CoV-2 у субъекта.
Прежде чем излагать это изобретение более подробно, может быть полезно для его понимания дать определения некоторых терминов, которые будут использоваться в настоящем документе. Дополнительные определения представлены на протяжении всего данного описания изобретения.
В контексте настоящего документа "SARS-CoV-2", также называемый в настоящем документе "уханьский коронавирус" или "уханьский вирус пневмонии рынка морепродуктов", или "уханьский CoV", или "новый CoV", или "nCoV", или "2019 nCoV", или "уханьский nCoV", представляет собой бетакоронавирус, предположительно принадлежащий к роду B (сарбековирус). SARS-CoV-2 был впервые выявлен в Ухане, провинция Хубэй, Китай, в конце 2019 года и распространился в Китае и других частях мира к началу 2020 года. Симптомы SARS-CoV-2 включают лихорадку, сухой кашель и одышку.
Геномная последовательность изолята SARS-CoV-2 Wuhan-Hu-1 представлена в SEQ ID NO:163 (см. также GenBank MN908947.3, 23.01.2020), а аминокислотная трансляция генома представлена в SEQ ID NO:164 (см. также GenBank QHD43416.1, 23.01.2020). Как и другие коронавирусы (например, SARS CoV), SARS-CoV-2 содержит трансмембранный гликопротеин I типа "спайк" или поверхностный ("S"), содержащий рецептор-связывающий домен (RBD). Считается, что RBD опосредует проникновение коронавируса SARS линии B в клетки эпителия дыхательных путей путем связывания с ангиотензинпревращающим ферментом 2 (ACE2). В частности, считается, что рецептор-связывающий мотив (RBM) в RBD вируса взаимодействует с ACE2.
Аминокислотная последовательность поверхностного гликопротеина (S) SARS-CoV-2 Wuhan-Hu-1 представлена в SEQ ID NO:165. Антитела и антигенсвязывающие фрагменты по настоящему изобретению способны связываться с поверхностным гликопротеином (S) SARS CoV-2, таким как Wuhan-Hu-1. Например, в некоторых вариантах осуществления антитело или антигенсвязывающий фрагмент связывается с эпитопом в RBD S-белка Wuhan-Hu-1.
Аминокислотная последовательность RBD SARS-CoV-2 Wuhan-Hu-1 представлена в SEQ ID NO:166. S-белок SARS-CoV-2 Wuhan-Hu-1 имеет приблизительно 73% идентичности с аминокислотной последовательностью с S-белком SARS-CoV. Аминокислотная последовательность RBM SARS-CoV-2 Wuhan-Hu-1 представлена в SEQ ID NO:167. SARS-CoV-2 RBD имеет сходство аминокислотной последовательности приблизительно от 75 до 77% с RBD коронавируса SARS, а SARS-CoV-2 Wuhan Hu-1RBM имеет сходство аминокислотной последовательности приблизительно 50% с RBM коронавируса SARS.
Если в настоящем документе не указано иное, SARS-CoV-2 Wuhan Hu-1 относится к вирусу, содержащему аминокислотную последовательность, представленную в любой одной или более из SEQ ID NO:164, 165 и 166, необязательно с геномной последовательностью, представленной в SEQ ID NO:163.
Существует ряд новых вариантов SARS-CoV-2. Некоторые варианты SARS-CoV-2 содержат мутацию N439K, которая обладает повышенной аффинностью связывания с рецептором ACE2 человека (Thomson, E.C., и др., The circulating SARS-CoV-2 spike variant N439K maintains fitness while evading antibody-mediated immunity. bioRxiv, 2020). Некоторые варианты SARS-CoV-2 содержат мутацию N501Y, которая связана с повышенной трансмиссивностью, включая линии B.1.1.7 (также известные как 20I/501Y.V1 и VOC 202012/01; (мутации del69-70, del144, N501Y, A570D, D614G, P681H, T716I, S982A, и D1118H)) и B.1.351 (также известные как 20H/501Y.V2; мутации L18F, D80A, D215G, R246I, K417N, E484K, N501Y, D614G и A701V), которые были обнаружены в Великобритании и Южной Африке, соответственно (Tegally,H., и др. al, Emergence and rapid spread of a new severe acute respiratory syndrome-related coronavirus 2 (SARS-CoV-2) lineage with multiple spike mutations in South Africa. medRxiv, 2020: p. 2020.12.21.2024864; Leung, K., и др., Early empirical assessment of the N501Y mutant strains of SARS-CoV-2 in the United Kingdom, October to November 2020. medRxiv, 2020: p. 2020.12.20.20248581). B.1.351 также содержит две другие мутации в домене RBD спайк-белка SARS-CoV2, K417N и E484K (Tegally, H., и др., Emergence and rapid spread of a new severe acute respiratory syndrome-related coronavirus 2 (SARS-CoV-2) lineage with multiple spike mutations in South Africa. medRxiv, 2020: p. 2020.12.21.202486400). Другие варианты SARS-CoV-2 включают линию B.1.1.28, которая была впервые зарегистрирована в Бразилии; вариант P.1, линия B.1.1.28 (также известная как 20J/501Y.V3), который был впервые зарегистрирован в Японии; вариант L452R, который был впервые зарегистрирован в Калифорнии в Соединенных Штатах (Pan American Health Organization, Epidemiological update: Occurrence of variants of SARS-CoV-2 in the Americas, January 20, 2021, доступно на reliefweb.int/sites/reliefweb.int/files/resources/2021-jan-20-phe-epi-update-SARS-CoV-2.pdf). Другие варианты SARS-CoV-2 включают SARS CoV-2 из группы 19A; SARS CoV-2 из группы 19B; SARS CoV-2 из группы 20A; SARS CoV-2 из группы 20B; SARS CoV-2 из группы 20C; SARS CoV-2 из группы 20D; SARS CoV-2 из группы 20E (EU1); SARS CoV-2 из группы 20F; SARS CoV-2 из группы 20G; и SARS CoV-2 B1.1.207; и другие линии SARS CoV-2, описанные в Rambaut, A., и др.., A dynamic nomenclature proposal for SARS-CoV-2 lineages to assist genomic epidemiology. Nat Microbiol 5, 1403-1407 (2020). Вышеуказанные варианты SARS-CoV-2 и их аминокислотные и нуклеотидные последовательности включены в настоящий документ посредством ссылки.
В настоящем описании любой диапазон концентраций, процентный диапазон, диапазон отношений или целочисленный диапазон, приведенные в настоящем документе, следует понимать как включающие значение любого целого числа в пределах указанного диапазона и, когда это необходимо, его фракций (таких как одна десятая и одна сотая целое число), если не указано иное. Кроме того, любой диапазон чисел, указанных в настоящем документе, относящийся к любому физическому признаку, такому как полимерные субъединицы, размер или толщина, следует понимать как включающее любое целое число в пределах указанного диапазона, если не указано иное. В контексте настоящего документа «около» означает ±20% от указанного диапазона, значения или структуры, если не указано иное. Следует понимать, что термины в единственном числе в контексте настоящего документа подразумевают "один или более" из перечисленных терминов. Таким образом, использование альтернативы (например, «или») следует понимать как одну, обе или любую комбинацию этих альтернатив. В контексте настоящего документа "включать", "иметь" и "содержать" используются как синонимы, а эти термины и их варианты следует рассматривать как неограничивающие.
«Необязательный» или «необязательно» означает, что описанный далее элемент, компонент, событие или обстоятельство необязательно должно иметь место, и что описание включает случаи, в которых элемент, компонент, событие или обстоятельство имеет место, и случаи, когда их нет.
Кроме того, следует понимать, что отдельные конструкции или группы конструкций, полученные из различных комбинаций структур и субъединиц, описанных в настоящей заявке, раскрыты в настоящей заявке в той же степени, как если бы каждая конструкция или группа конструкций была изложена отдельно. Таким образом, выбор конкретных структур или конкретных субъединиц находится в пределах объема настоящего изобретения.
Термин «состоящий по существу из» не эквивалентен термину «содержащий» и относится к указанным материалам или стадиям формулы изобретения или к тем, которые не оказывают существенного влияния на основные характеристики заявленного объекта изобретения. Например, белковый домен, область или модуль (например, связывающий домен) или белок "состоит по существу из" конкретной аминокислотной последовательности, когда аминокислотная последовательность домена, области, модуля или белка включает удлинения, делеции, мутации или их комбинацию (например, аминокислоты на амино- или карбоксиконце или между доменами), которые в комбинации вносят вклад не более чем в 20% (например, не более 15%, 10%, 8%, 6%, 5%, 4%, 3%, 2% или 1%) длины домена, области, модуля или белка и по существу не влияют (т.е., не снижают активность более чем на 50%, например, не более чем на 40%, 30%, 25%, 20%, 15%, 10%, 5% или 1%) на активность домена (доменов), области (областей), модуля (модулей) или белка (например, на аффинность связывания с мишенью связывающего белка).
В контексте настоящего документа "аминокислота" относится к встречающимся в природе и синтетическим аминокислотам, а также аналогам аминокислот и миметикам аминокислот, которые функционируют аналогично встречающимся в природе аминокислотам. Природные аминокислоты представляют собой аминокислоты, кодируемые генетическим кодом, а также те аминокислоты, которые впоследствии модифицируются, например, гидроксипролин, γ-карбоксиглутамат и O-фосфосерин. Аналоги аминокислот относятся к соединениям, которые имеют ту же основную химическую структуру, что и встречающаяся в природе аминокислота, то есть, α-углерод, который связан с водородом, карбоксильной группой, аминогруппой и R-группой, например, гомосерин, норлейцин, метионинсульфоксид, метионинметилсульфоний. Такие аналоги имеют модифицированные R-группы (например, норлейцин) или модифицированные пептидные остовы, но сохраняют ту же основную химическую структуру, что и встречающаяся в природе аминокислота. Миметики аминокислот относятся к химическим соединениям, которые имеют структуру, которая отличается от общей химической структуры аминокислоты, но которая функционирует аналогично встречающейся в природе аминокислоте.
В контексте настоящего документа "мутация" относится к изменению последовательности молекулы нуклеиновой кислоты или молекулы полипептида по сравнению с эталонной молекулой или молекулой нуклеиновой кислоты дикого типа или молекулой полипептида, соответственно. Мутация может привести к нескольким различным типам изменения последовательности, включая замену, вставку или делецию нуклеотида (нуклеотидов) или аминокислоты (аминокислот).
"Консервативная замена" относится к аминокислотным заменам, которые существенно не влияют или не изменяют характеристики связывания конкретного белка. Как правило, консервативные замены представляют собой замены, в которых замещенный аминокислотный остаток заменен аминокислотным остатком, имеющим схожую боковую цепь. Консервативные замены включают замену, основанную на одной из следующих групп: группа 1: аланин (Ala или A), глицин (Gly или G), серин (Ser или S), треонин (Thr или T); группа 2: аспарагиновая кислота (Asp или D), глутаминовая кислота (Glu или Z); группа 3: аспарагин (Asn или N), глутамин (Gln или Q); группа 4: аргинин (Arg или R), лизин (Lys или K), гистидин (His или H); группа 5: изолейцин (Ile или I), лейцин (Leu или L), метионин (Met или M), валин (Val или V); и группа 6: фенилаланин (Phe или F), тирозин (Tyr или Y), триптофан (Trp или W). Дополнительно или альтернативно, аминокислоты могут быть сгруппированы в консервативные группы замещения по аналогичной функции, химической структуре или составу (например, кислотные, основные, алифатические, ароматические или серосодержащие). Например, алифатическая группа может включать, для целей замены, Gly, Ala, Val, Leu и Ile. Другие группы консервативных замен включают: серосодержащие: Met и цистеин (Cys или C); кислые: Asp, Glu, Asn и Gln; небольшие алифатические, неполярные или слегка полярные остатки: Ala, Ser, Thr, Pro и Gly; полярные, отрицательно заряженные остатки и их амиды: Asp, Asn, Glu и Gln; полярные, положительно заряженные остатки: His, Arg и Lys; большие алифатические, неполярные остатки: Met, Leu, Ile, Val и Cys; и большие ароматические остатки: Phe, Tyr и Trp. Дополнительную информацию можно найти в Creighton (1984) Proteins, W.H. Freeman and Company.
В контексте настоящего документа "белок" или "полипептид" относится к полимеру аминокислотных остатков. Белки применяются к встречающимся в природе аминокислотным полимерам, а также к аминокислотным полимерам, в которых один или более аминокислотных остатков являются искусственными химическими миметиками соответствующей встречающейся в природе аминокислоты, и не встречающимся в природе аминокислотным полимерам. Также рассматриваются варианты белков, пептидов и полипептидов согласно настоящему изобретению. В некоторых вариантах осуществления варианты белков, пептидов и полипептидов содержат или состоят из аминокислотной последовательности, которая по меньшей мере на 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 99,9% идентична аминокислотной последовательности определенной или эталонной аминокислотной последовательности, как описано в настоящем документе.
"Молекула нуклеиновой кислоты" или "полинуклеотид" или "полинуклеиновая кислота" относится к полимерному соединению, включающему ковалентно связанные нуклеотиды, которые могут состоять из природных субъединиц (например, пуриновые или пиримидиновые основания) или неприродных субъединиц (например, морфолиновое кольцо). Пуриновые основания включают аденин, гуанин, гипоксантин и ксантин, а пиримидиновые основания включают урацил, тимин и цитозин. Молекулы нуклеиновой кислоты включают полирибонуклеиновую кислоту (РНК), которая включает мРНК (матричная РНК), микроРНК, миРНК (малая интерферирующая РНК), вирусную геномную РНК и синтетическую РНК, и полидезоксирибонуклеиновую кислоту (ДНК), которая включает кДНК (комплементарная ДНК), геномную ДНК и синтетическую ДНК, каждая из которых может быть одноцепочечной или двухцепочечной. В случае одноцепочечной цепи молекула нуклеиновой кислоты может представлять собой кодирующую цепь или некодирующую (антисмысловую) цепь. Молекула нуклеиновой кислоты, кодирующая аминокислотную последовательность, включает все нуклеотидные последовательности, кодирующие одну и ту же аминокислотную последовательность. Некоторые варианты нуклеотидных последовательностей могут также включать интрон (интроны) в той степени, в которой интрон (интроны) будет удален с помощью ко- или посттранскрипционных механизмов. Другими словами, различные нуклеотидные последовательности могут кодировать одну и ту же аминокислотную последовательность в результате избыточности или вырожденности генетического кода или путем сплайсинга.
Также рассматриваются варианты молекул нуклеиновых кислот по данному описанию. Варианты молекул нуклеиновой кислоты имеют по меньшей мере 70%, 75%, 80%, 85%, 90% и предпочтительно на 95%, 96%, 97%, 98%, 99% или 99,9% идентичности с молекулой нуклеиновой кислоты определенного или эталонного полинуклеотида, как описано в настоящем документе, или которые гибридизуются с полинуклеотидом в жестких условиях гибридизации, составляющих 0,015 М хлорида натрия, 0,0015 М цитрата натрия при около 65-68 °C или 0,015 М хлорида натрия, 0,0015 М цитрата натрия и 50% формамида при около 42 °C. Варианты молекул нуклеиновых кислот сохраняют способность кодировать их связывающий домен, имеющий функциональность, описанную в настоящем документе, такую как связывание молекулы-мишени.
"Процент идентичности последовательностей" относится к взаимосвязи между двумя или более последовательностями, как определено путем сравнения последовательностей. Предпочтительные способы определения идентичности последовательностей предназначены для обеспечения наилучшего соответствия между сравниваемыми последовательностями. Например, последовательности выровнены для целей оптимального сравнения (например, гэпы могут быть введены в одну или обе из первой и второй аминокислотной или нуклеотидной последовательности для оптимального выравнивания). Кроме того, негомологичные последовательности могут быть проигнорированы для целей сравнения. Процент идентичности последовательности, указанный в настоящем документе, рассчитывают по длине эталонной последовательности, если не указано иное. Способы определения идентичности и сходства последовательностей можно найти в общедоступных компьютерных программах. Выравнивание последовательностей и расчеты процента идентичности могут быть выполнены с использованием программы BLAST (например, BLAST 2.0, BLASTP, BLASTN или BLASTX). Математический алгоритм, используемый в программах BLAST, можно найти в Altschul и др., Nucleic Acids Res. 25:3389-3402, 1997. В контексте настоящего документа следует понимать, что для анализа при применении аналитического программного обеспечения результаты анализа основаны на «установленных по умолчанию значениях» упоминаемой программы. «Установленные по умолчанию значения» означают любой набор значений или показателей, загружаемых исходно с программным обеспечением при первом запуске.
Термин «выделенный» означает, что материал извлечен из своего исходного окружения (например, природной среды, если он встречается в естественных условиях). Например, встречающаяся в природе нуклеиновая кислота или полипептид, присутствующий в живом животном, не является выделенным, но та же нуклеиновая кислота или полипептид, отделенный от материалов, совместно с ним присутствующих в естественной системе, является выделенным. Такая нуклеиновая кислота может быть частью вектора и/или такая нуклеиновая кислота или полипептид могут быть частью композиции (например, клеточного лизата) и все еще могут быть выделены в том смысле, что такой вектор или композиция не является частью природной среды для нуклеиновой кислоты или полипептида. "Выделенный" может, в некоторых вариантах осуществления, также описывать антитело, антигенсвязывающий фрагмент, полинуклеотид, вектор, клетку-хозяина или композицию, которая находится вне организма человека.
Термин "ген" означает сегмент ДНК или РНК, участвующий в получении полипептидной цепи; в некоторых контекстах он включает области, предшествующие и следующие за кодирующей областью (например, 5’-нетранслируемая область (UTR) и 3’-UTR), а также промежуточные последовательности (интроны) между отдельными кодирующими сегментами (экзонами).
"Функциональный вариант" относится к полипептиду или полинуклеотиду, который структурно схож или по существу структурно схож с исходным или эталонным соединением согласно настоящему изобретению, но незначительно отличается по составу (например, одно основание, атом или функциональная группа отличаются, добавлены или удалены), так что полипептид или кодируемый полипептид способен выполнять по меньшей мере одну функцию исходного полипептида с по меньшей мере 50% эффективностью, предпочтительно по меньшей мере 55%, 60%, 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99%, 99,9% или 100% уровнем активности исходного полипептида. Другими словами, функциональный вариант полипептида или кодируемого полипептида согласно настоящему изобретению имеет "сходное связывание", "сходную аффинность" или "сходную активность", когда функциональный вариант демонстрирует снижение производительности в выбранном анализе не более чем на 50% по сравнению с исходным или эталонным полипептидом, таким как анализ для измерения аффинности связывания (например, окрашивание Biacore® или тетрамером, измеряющее константу ассоциации (Ka) или диссоциации (KD)).
В контексте настоящего документа "функциональная часть" или "функциональный фрагмент" относится к полипептиду или полинуклеотиду, который содержит только домен, часть или фрагмент исходного или эталонного соединения, и полипептид или кодируемый полипептид сохраняет по меньшей мере 50% активность, связанную с доменом, частью или фрагментом исходного или эталонного соединения, предпочтительно по меньшей мере 55%, 60%, 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99%, 99,9% или 100% уровень активности исходного полипептида, или обеспечивает биологическое преимущество (например, эффекторную функцию). «Функциональная часть» или «функциональный фрагмент» полипептида или кодируемого полипептида согласно настоящему изобретению имеет «аналогичное связывание» или «аналогичную активность», когда функциональная часть или фрагмент демонстрирует снижение производительности в выбранном анализе не более чем на 50% по сравнению с исходным или эталонным полипептидом (предпочтительно не более чем на 20% или 10%, или не более чем на логарифмическую (log) разницу по сравнению с исходным или эталонным по отношению к аффинности).
В контексте настоящего документа термин "сконструированный", "рекомбинантный" или "неприродный" относится к организму, микроорганизму, клетке, молекуле нуклеиновой кислоты или вектору, который включает по меньшей мере одно генетическое изменение или был модифицирован путем введения экзогенной или гетерологичной молекулы нуклеиновой кислоты, где такие изменения или модификации вводятся с помощью генной инженерии (т.е.вмешательства человека). Генетические изменения включают, например, модификации, вводящие экспрессируемые молекулы нуклеиновых кислот, кодирующие функциональную РНК, белки, слитые белки или ферменты, или другие добавления, делеции, замены молекул нуклеиновых кислот или другие функциональные нарушения генетического материала клетки. Дополнительные модификации включают, например, некодирующие регуляторные области, в которых модификации изменяют экспрессию полинуклеотида, гена или оперона.
В контексте настоящего документа термин "гетерологичный" или "неэндогенный" или "экзогенный" относится к любому гену, белку, соединению, молекуле нуклеиновой кислоты или активности, которая не является нативной для клетки-хозяина или субъекта, или любому гену, белку, соединению, молекуле нуклеиновой кислоты или активности, которая является нативной для клетки-хозяина или субъекта, который был изменен. Гетерологичные, неэндогенные или экзогенные включают гены, белки, соединения или молекулы нуклеиновых кислот, которые были мутированы или иным образом изменены таким образом, что структура, активность или и то и другое различаются между нативными и измененными генами, белками, соединениями или молекулами нуклеиновых кислот. В некоторых вариантах осуществления гетерологичные, неэндогенные или экзогенные гены, белки или молекулы нуклеиновых кислот (например, рецепторы, лиганды и т.д.) могут быть неэндогенными для клетки-хозяина или субъекта, но вместо этого нуклеиновые кислоты, кодирующие такие гены, белки или молекулы нуклеиновых кислот, могут быть добавлены в клетку-хозяина путем конъюгации, трансформации, трансфекции, электропорации или тому подобного, где добавленная молекула нуклеиновой кислоты может интегрироваться в геном клетки-хозяина или может существовать в виде внехромосомного генетического материала (например, в виде плазмиды или другого самовоспроизводящегося вектора). Термин "гомологичный" или "гомолог" относится к гену, белку, соединению, молекуле нуклеиновой кислоты или активности, обнаруженной в или полученной из клетки-хозяина, вида или штамма. Например, гетерологичный или экзогенный полинуклеотид или ген, кодирующий полипептид, может быть гомологичным нативному полинуклеотиду или гену и кодировать гомологичный полипептид или активность, но полинуклеотид или полипептид может иметь измененную структуру, последовательность, уровень экспрессии или любую их комбинацию. Неэндогенный полинуклеотид или ген, а также кодируемый полипептид или активность могут быть из одного и того же вида, другого вида или их комбинации.
В некоторых вариантах осуществления молекула нуклеиновой кислоты или ее часть, нативная для клетки-хозяина, будет считаться гетерологичной по отношению к клетке-хозяину, если она была изменена или мутирована, или молекула нуклеиновой кислоты, нативная для клетки-хозяина, может считаться гетерологичной, если она была изменена с помощью гетерологичной последовательности контроля экспрессии или была изменена с помощью эндогенной последовательности контроля экспрессии, обычно не связанной с молекулой нуклеиновой кислоты, нативной для клетки-хозяина. Кроме того, термин "гетерологичный" может относиться к биологической активности, которая отличается, изменена или не эндогенна для клетки-хозяина. Как описано в настоящем документе, более чем одна гетерологичная молекула нуклеиновой кислоты может быть введена в клетку-хозяина в виде отдельных молекул нуклеиновой кислоты, в виде множества индивидуально контролируемых генов, в виде молекулы полицистронной нуклеиновой кислоты, в виде одной молекулы нуклеиновой кислоты, кодирующей антитело или антигенсвязывающий фрагмент (или другой полипептид), или любую их комбинацию.
В контексте настоящего документа термин "эндогенный" или "нативный" относится к полинуклеотиду, гену, белку, соединению, молекуле или активности, которые обычно присутствуют в клетке-хозяине или субъекте.
В контексте настоящего документа «экспрессия» в данном контексте относится к процессу, посредством которого получали полипептид на основе кодирующей последовательности молекулы нуклеиновой кислоты, такой как ген. Способ может включать транскрипцию, посттранскрипционный контроль, посттранскрипционную модификацию, трансляцию, посттрансляционный контроль, посттрансляционную модификацию или любую их комбинацию. Экспрессируемая молекула нуклеиновой кислоты обычно функционально связана с последовательностью контроля экспрессии (например, промотором).
Термин "функционально связанный" относится к ассоциации двух или более молекул нуклеиновой кислоты на одном фрагменте нуклеиновой кислоты, так что функция одной из них зависит от другой. Например, промотор функционально связан с кодирующей последовательностью, если он способен регулировать экспрессию указанной кодирующей последовательности (т.е. кодирующая последовательность находится под транскрипционным контролем указанного промотора). "Несвязанный" означает, что ассоциированные генетические элементы не тесно связаны друг с другом и функция одного не влияет на другого.
Как описано в настоящем документе, более чем одна гетерологичная молекула нуклеиновой кислоты может быть введена в клетку-хозяина в виде отдельных молекул нуклеиновой кислоты, в виде множества индивидуально контролируемых генов, в виде молекулы полицистронной нуклеиновой кислоты, в виде одной молекулы нуклеиновой кислоты, кодирующей белок (например, тяжелую цепь антитела) или любую их комбинацию. Когда в клетку-хозяина вводят две или более гетерологичных молекул нуклеиновой кислоты, следует понимать, что две или более гетерологичных молекул нуклеиновой кислоты могут быть введены в виде одной молекулы нуклеиновой кислоты (например, на одном векторе), на отдельных векторах, интегрированных в хромосому-хозяина на одном сайте или нескольких сайтах, или любой их комбинации. Количество упомянутых гетерологичных молекул нуклеиновых кислот или белковых активностей относится к количеству кодирующих молекул нуклеиновых кислот или количеству белковых активностей, а не к количеству отдельных молекул нуклеиновых кислот, введенных в клетку-хозяина.
Термин «конструкция» относится к любому полинуклеотиду, который содержит рекомбинантную молекулу нуклеиновой кислоты (или, когда контекст явно указывает, слитый белок по настоящему изобретению). Конструкция (полинуклеотида) может присутствовать в векторе (например, бактериальном векторе, вирусном векторе) или может быть интегрирована в геном. "Вектор" представляет собой молекулу нуклеиновой кислоты, которая способна транспортировать другую молекулу нуклеиновой кислоты. Векторы могут представлять собой, например, плазмиды, космиды, вирусы, вектор РНК или линейную или круговую молекулу ДНК или РНК, которые могут включать хромосомные, нехромосомные, полусинтетические или синтетические молекулы нуклеиновых кислот. Векторы согласно настоящему изобретению также включают транспозонные системы (например, Sleeping Beauty, см., например, Geurts и др., Mol. Ther. 8:108, 2003: Mátés и др., Nat. Genet. 41:753, 2009). Примерные векторы представляют собой векторы, способные к автономной репликации (эписомальный вектор), способные доставлять полинуклеотид в геном клетки (например, вирусный вектор) или способные экспрессировать молекулы нуклеиновых кислот, с которыми они связаны (экспрессионные векторы).
В контексте настоящего документа "вектор экспрессии" или "вектор" относится к конструкции ДНК, содержащей молекулу нуклеиновой кислоты, которая функционально связана с подходящей контрольной последовательностью, способной осуществлять экспрессию молекулы нуклеиновой кислоты в подходящем хозяине. Такие контрольные последовательности включают промотор для осуществления транскрипции, необязательную операторную последовательность для контроля такой транскрипции, последовательность, кодирующую подходящие сайты связывания мРНК-рибосом, и последовательности, которые контролируют терминацию транскрипции и трансляции. Вектор может представлять собой плазмиду, фаговую частицу, вирус или просто потенциальную геномную вставку. После трансформации в подходящего хозяина вектор может реплицироваться и функционировать независимо от генома хозяина, или в некоторых случаях может интегрироваться в сам геном или доставлять полинуклеотид, содержащийся в векторе, в геном без последовательности вектора. В настоящем описании термины "плазмида", "экспрессионная плазмида", "вирус" и "вектор" часто используются взаимозаменяемо.
Термин "вводимый" в контексте вставки молекулы нуклеиновой кислоты в клетку означает "трансфекцию", "трансформацию" или "трансдукцию" и включает ссылку на включение молекулы нуклеиновой кислоты в эукариотическую или прокариотическую клетку, где молекула нуклеиновой кислоты может быть включена в геном клетки (например, хромосомы, плазмиды, пластиды или митохондриальной ДНК), преобразована в автономны репликон или временно экспрессирована (например, трансфицированная мРНК).
В некоторых вариантах осуществления полинуклеотиды согласно настоящему изобретению могут быть функционально связаны с определенными элементами вектора. Например, полинуклеотидные последовательности, которые необходимы для осуществления экспрессии и процессинга кодирующих последовательностей, с которыми они лигированы, могут быть функционально связаны. Последовательности контроля экспрессии могут включать соответствующие последовательности инициации, терминации, промотора и энхансера транскрипции; эффективные сигналы процессинга РНК, такие как сигналы сплайсинга и полиаденилирования; последовательности, которые стабилизируют цитоплазматическую мРНК; последовательности, которые повышают эффективность трансляции (т.е. консенсусные последовательности Козака); последовательности, которые повышают стабильность белка; и, возможно, последовательности, которые повышают секрецию белка. Последовательности контроля экспрессии могут быть функционально связаны, если они смежны с представляющим интерес геном и последовательностями контроля экспрессии, которые действуют в транс или на расстоянии для контроля представляющего интерес гена.
В некоторых вариантах осуществления вектор содержит плазмидный вектор или вирусный вектор (например, лентивирусный вектор или γ-ретровирусный вектор). Вирусные векторы включают ретровирус, аденовирус, парвовирус (например, аденоассоциированные вирусы), коронавирус, вирусы РНК с отрицательной цепью, такие как ортомиксовирус (например, вирус гриппа), рабдовирус (например, вирус бешенства и везикулярного стоматита), парамиксовирус (например, корь и Сендай), вирусы РНК с положительной цепью, такие как пикорнавирус и альфавирус, и вирусы двухцепочечной ДНК, включая аденовирус, герпесвирус (например, вирус простого герпеса 1 и 2 типов, вирус Эпштейна-Барра, цитомегаловирус) и поксвирус (например, осповакцны, оспу кур и оспу канареек). Другие вирусы включают, например, вирус Норуолк, тогавирус, флавивирус, реовирусы, паповавирус, гепаднавирус и вирус гепатита. Примеры ретровирусов включают лейкоз-саркому птиц, вирусы C-типа млекопитающих, вирусы B-типа, вирусы D-типа, группу HTLV-BLV, лентивирус, спумавирус (Coffin, J. M., Retroviridae: The viruses and their replication, In Fundamental Virology, Third Edition, B. N. Fields et al., Eds., Lippincott-Raven Publishers, Philadelphia, 1996).
"Ретровирусы" представляют собой вирусы, имеющие геном РНК, который обратно транскрибируется в ДНК с использованием фермента обратной транскриптазы, затем обратно транскрибируемая ДНК включается в геном клетки-хозяина. "Gammaretrovirus" относится к роду ретровирусов (Retroviridae). Примеры гаммаретровирусов включают вирус стволовых клеток мыши, вирус лейкоза мыши, вирус лейкоза кошек, вирус саркомы кошек и вирусы ретикулоэндотелиоза птиц.
"Лентивирусные векторы" включают в себя основанные на ВИЧ (вирус иммунодефицита человека) лентивирусные векторы для доставки генов, которые могут быть интегрирующими или неинтегрирующими, обладают относительно большой упаковочной емкостью и могут трансдуцировать ряд различных типов клеток. Лентивирусные векторы обычно генерируют после временной трансфекции трех (упаковки, оболочки и переноса) или более плазмид в клетки-продуценты. Как и ВИЧ, лентивирусные векторы проникают в клетку-мишень через взаимодействие гликопротеинов вирусной поверхности с рецепторами на поверхности клетки. При вводе вирусная РНК подвергается обратной транскрипции, которая опосредуется комплексом вирусной обратной транскриптазы. Продуктом обратной транскрипции является двухцепочечная линейная вирусная ДНК, которая является субстратом для вирусной интеграции в ДНК инфицированных клеток.
В некоторых вариантах осуществления вирусный вектор может представлять собой гаммаретровирус, например, векторы, полученные из вируса лейкоза мышей Молони (MLV). В других вариантах осуществления вирусный вектор может представлять собой более сложный вектор, полученный из ретровируса, например, вектор, полученный из лентивируса. К этой категории относятся векторы, происходящие из ВИЧ-1. Другие примеры включают лентивирусные векторы, полученные из ВИЧ-2, FIV (вирус иммунодефицита кошки), вируса инфекционной анемии лошадей, SIV и вируса Маеди-Висна (лентивируса овец). Способы применения ретровирусных и лентивирусных вирусных векторов и упаковки клеток для трансдукции клеток-хозяев млекопитающих с вирусными частицами, содержащими трансгены, известны в данной области техники и были описаны ранее, например, в: патенте США 8119772; Walchli и др., PLoS One 6:327930, 2011; Zhao и др., J. Immunol. 174:4415, 2005; Engels et al., Hum. Gene Ther. 14:1155, 2003; Frecha и др., Mol. Ther.18:1748, 2010; и Verhoeyen и др., Methods Mol. Biol. 506:97, 2009. Ретровирусные и лентивирусные векторные конструкции и системы экспрессии также коммерчески доступны. Другие вирусные векторы также могут быть использованы для доставки полинуклеотидов, включая ДНК-вирусные векторы, включая, например, векторы на основе аденовируса и векторы на основе аденоассоциированного вируса (AAV); векторы, полученные из вирусов простого герпеса (HSV), включая векторы ампликона, репликационно дефектный HSV и аттенуированный HSV (Krisky и др., Gene Ther. 5:1517, 1998).
Другие векторы, которые могут быть использованы с композициями и способами согласно настоящему изобретению, включают векторы, полученные из бакуловирусов и α-вирусов. (Jolly, D J. 1999. Emerging Viral Vectors. pp. 209-40 in Friedmann T. ed. The Development of Human Gene Therapy. New York: Cold Spring Harbor Lab), или плазмидные векторы (такие как "спящая красавица" или другие транспозонные векторы).
Когда геном вирусного вектора содержит множество полинуклеотидов, подлежащих экспрессии в клетке-хозяине в виде отдельных транскриптов, вирусный вектор может также содержать дополнительные последовательности между двумя (или более) транскриптами, обеспечивающие бицистронную или мультицистронную экспрессию. Примеры таких последовательностей, используемых в вирусных векторах, включают внутренние сайты входа рибосом (IRES), сайты расщепления фурином, вирусный пептид 2А или любую их комбинацию.
Плазмидные векторы, включая плазмидные векторы, кодирующие антитело на основе ДНК или антигенсвязывающий фрагмент, для прямого введения субъекту, описаны далее в настоящем документе.
В данном контексте термин "хозяин" относится к клетке или микроорганизму, предназначенному для генетической модификации гетерологичной молекулой нуклеиновой кислоты с получением представляющего интерес полипептида (например, антитела по настоящему изобретению).
Клетка-хозяин может включать любую отдельную клетку или клеточную культуру, которая может получать вектор или включение нуклеиновых кислот или экспрессирующих белков. Термин также охватывает потомство клетки-хозяина, генетически или фенотипически одинаковое или различное. Подходящие клетки-хозяева могут зависеть от вектора и могут включать клетки млекопитающих, клетки животных, клетки человека, клетки обезьяны, клетки насекомых, клетки дрожжей и бактериальные клетки. Эти клетки могут быть индуцированы для включения вектора или другого веества с помощью вирусного вектора, трансформации путем осаждения фосфата кальция, DEAE-декстрана (DEAE - диэтиламиноэтил), электропорации, микроинъекции или других способов. См., например, Sambrook и др., Molecular Cloning: A Laboratory Manual 2d ed. (Cold Spring Harbor Laboratory, 1989).
В контексте инфекции SARS-CoV-2 "хозяин" относится к клетке или субъекту (например, человеку), инфицированному SARS-CoV-2.
"Антиген" или "Ag" в данном контексте относится к иммуногенной молекуле, которая вызывает иммунный ответ. Этот иммунный ответ может включать продукцию антител, активацию специфических иммунологически-компетентных клеток, активацию комплемента, антителозависимую цитотоксичность или любую их комбинацию. Антиген (иммуногенная молекула) может представлять собой, например, пептид, гликопептид, полипептид, гликополипептид, полинуклеотид, полисахарид, липид или т. п. Очевидно, что антиген может быть синтезирован, получен рекомбинантным способом или получен из биологического образца. Примерные биологические образцы, которые могут содержать один или более антигенов, включают образцы тканей, образцы кала, клетки, биологические жидкости или их комбинации. Антигены могут продуцироваться клетками, которые были модифицированы или генетически сконструированы для экспрессии антигена. Антигены также могут присутствовать в SARS-CoV-2 (например, поверхностном гликопротеине или его части), например, присутствовать в вирионе, или экспрессироваться или презентироваться на поверхности клетки, инфицированной SARS-CoV-2.
Термин "эпитоп" или "антигенный эпитоп" включает любую молекулу, структуру, аминокислотную последовательность или детерминанту белка, которая распознается и специфически связывается родственной связывающей молекулой, такой как иммуноглобулин, или другой связывающей молекулой, доменом или белком. Эпитопные детерминанты обычно содержат расположенные на химически активные поверхностные группы молекул, такие как боковые цепи аминокислот или сахаров, и обычно обладают специфическими трехмерными структурными характеристиками, а также специфическими характеристиками заряда. Если антиген представляет собой или содержит пептид или белок, эпитоп может состоять из последовательных аминокислот (например, линейного эпитопа) или может состоять из аминокислот из различных частей или областей белка, которые сближены за счет сворачивания белка (например, прерывистого или конформационного эпитопа), или несмежных аминокислот, которые находятся в непосредственной близости независимо от сворачивания белка.
Антитела и антигенсвязывающие фрагменты
В одном аспекте настоящее изобретение относится к выделенному антителу или его антигенсвязывающему фрагменту, которые содержат вариабельный домен тяжелой цепи (VH), содержащий CDRH1, CDRH2 и CDRH3, и вариабельный домен легкой цепи (VL), содержащий CDRL1, CDRL2 и CDRL3, и способны связываться с поверхностным гликопротеином (S) SARS-CoV-2. В некоторых вариантах осуществления антитело или антигенсвязывающий фрагмент способно связываться с поверхностным гликопротеином (S) SARS-CoV-2, экспрессируемом на поверхности клетки-хозяина, и/или на вирионе SARS-CoV-2.
В некоторых вариантах осуществления антитело или антигенсвязывающий фрагмент по настоящему изобретению ассоциируется или объединяется с поверхностным эпитопом или антигеном гликопротеина SARS-CoV-2, содержащим указанный эпитоп, при этом существенно не ассоциируясь или не объединяясь с любыми другими молекулами или компонентами в образце.
В некоторых вариантах осуществления антитело или антигенсвязывающий фрагмент по настоящему изобретению связывается или объединяется (например, связывается) с поверхностным эпитопом гликопротеина SARS-CoV-2, а также может связываться или объединяться с эпитопом из другого коронавируса (например, SARS CoV), присутствующего в образце, но существенно не ассоциирующегося или объединяющегося с любыми другими молекулами или компонентами в образце. Другими словами, в некоторых вариантах осуществления антитело или антигенсвязывающий фрагмент по настоящему изобретению является перекрестно реактивным по отношению к SARS-CoV-2 и одному или более дополнительным коронавирусам.
В некоторых вариантах осуществления антитело или антигенсвязывающий фрагмент по настоящему изобретению специфически связывается с поверхностным гликопротеином SARS-CoV-2. В контексте настоящего документа "специфически связывается" относится к ассоциации или объединению антитела или антигенсвязывающего фрагмента с антигеном с аффинностью или Ka (т.е. константой равновесной ассоциации конкретного связывающего взаимодействия с единицами измерения 1/M) с 105 M-1 или более (что равно отношению скорости ассоциации [Kon] к скорости диссоциации [Koff] для этой реакции ассоциации), при этом не ассоциируясь или не объединяясь в значительной степени с любыми другими молекулами или компонентами в образце.В качестве альтернативы, аффинность может быть определена как равновесная константа диссоциации (Kd) отдельного связывающего взаимодействия, выражаемая в единицах измерения М (например, от 10-5 M до 10-13 M).Антитела могут быть классифицированы как "высокоаффинные" антитела или как "низкоаффинные" антитела. "Высокоаффинные" антитела относятся к тем антителам, которые имеют Ka по меньшей мере 107 M-1, по меньшей мере 108 M-1, по меньшей мере109 M-1, по меньшей мере 1010 M-1, по меньшей мере1011 M-1, по меньшей мере 1012 M-1или по меньшей мере 1013 M-1. «Низкоаффинные» антитела относятся к тем антителам, которые имеют Ka до 107 M-1, до 106 M-1, до 105 M-1. В качестве альтернативы, аффинность может быть определена как равновесная константа диссоциации (Kd) отдельного связывающего взаимодействия, выражаемая в единицах измерения М (например, от 10-5 M до 10-13 M).
В некоторых контекстах антитело и антигенсвязывающие фрагменты могут быть описаны со ссылкой на аффинность и/или авидность к антигену. Если не указано иное, авидность относится к общей силе связывания антитела или его антигенсвязывающего фрагмента с антигеном и отражает аффинность связывания, валентность антитела или антигенсвязывающего фрагмента (например, содержит ли антитело или антигенсвязывающий фрагмент один, два, три, четыре, пять, шесть, семь, восемь, девять, десять или более сайтов связывания) и, например, присутствует ли другой агент, который может влиять на связывание (например, неконкурентный ингибитор антитела или антигенсвязывающего фрагмента).
Известно множество анализов для идентификации антител по настоящему изобретению, которые связывают конкретную мишень, а также для определения аффинности связывающего домена или связывающего белка, таких как вестерн-блоттинг, ELISA (например, прямой, непрямой или сэндвич), аналитическое ультрацентрифугирование, спектроскопия и анализ поверхностного плазмонного резонанса (Biacore®) (см., например, Scatchard и др., Ann. N.Y. Acad. Sci. 51:660, 1949; Wilson, Science 295:2103, 2002; Wolff и др., Cancer Res. 53:2560, 1993; и патенты США № 5,283,173, 5,468,614 или их эквивалент). Также известны анализы для оценки аффинности или кажущейся аффинности или относительной аффинности.
В некоторых примерах связывание может быть определено путем рекомбинантной экспрессии антигена SARS-CoV-2 в клетке-хозяине (например, путем трансфекции) и иммуноокрашивания (например, фиксированной или фиксированной и пермеабилизированной) клетки-хозяина антителом и анализа связывания с помощью проточной цитометрии (например, с использованием анализатора клеток ZE5 (BioRad®) и программного обеспечения FlowJo (TreeStar). В некоторых вариантах осуществления положительное связывание может быть определено дифференциальным окрашиванием антителом клеток, экспрессирующих SARS-CoV-2, по сравнению с контрольными (например, имитирующими) клетками.
В некоторых вариантах осуществления антитело или антигенсвязывающий фрагмент по настоящему изобретению связывается с S-белком SARS-CoV-2, как измерено с помощью биослойной интерферометрии. В некоторых вариантах осуществления антитело или антигенсвязывающий фрагмент согласно настоящему изобретению связывается с S-белком SARS-CoV-2 с KD менее чем около 4,5×10-9 M, менее чем около 5×10-9 M, менее чем около 1×10-10 M, менее чем около 5×10-10 M, менее чем около 1×10-11 M, менее чем около 5×10-11 M, менее чем около 1×10-12 M или менее чем около 5×10-12 M. В некоторых вариантах осуществления антитело или антигенсвязывающий фрагмент по настоящему изобретению связывается с RBD S-белка SARS-CoV-2 с KD менее чем около 4,5×10-9 M, менее чем около 5×10-9 M, менее чем около 1×10-10 M, менее чем около 5×10-10 M, менее чем около 1×10-11M, менее чем около 5×10-11 M, менее чем около1×10-12 M или менее чем около 5×10-12 M. В некоторых вариантах осуществления антитело или антигенсвязывающий фрагмент по настоящему изобретению связывается с S-белком SARS-CoV-2 (например, гликозилированным или дегликозилированным S-белком RBD) с KD, ка и/или kd, как показано в таблице 8, таблице 9 или таблице 10 настоящего документа.
В частных вариантах осуществления антитело или его антигенсвязывающий фрагмент способно связываться с гликозилированным S-белком RBD с KD, составляющей около 0,35 нМ, около 0,36 нМ, около 0,37 нМ, около 0,38 нМ, около 0,39 нМ, около 0,40 нМ, около 0,41 нМ, около 0,42 нМ, около 0,43 нМ, около 0,44 нМ, около 0,45 нМ, около 0,46 нМ, около 0,47 нМ, около 0,48 нМ, около 0,49 нМ, около 0,50 нМ, около 0,51 нМ или около 1,7 нМ, необязательно измеренным с помощью поверхностного плазмонного резонанса, и/или с ka, составляющей около 8,5e4 1/Мс, около 8,6e4 1/Мс, около 8,7e4 1/Мс, около 8,8e4 1/Мс, около 8,9e4 1/Мс, около 9,0e4 1/Мс, около 9,1e4 1/Мс, около 9,2e4 1/Мс, около 9,3e4 1/Мс, около 9,4e4 1/Мс, около 9,5e4 1/Мс, около 9,6e4 1/Мс, около 9,7e4 1/Мс, около 9,8e4 1/Мс, около 9,9e4 1/Мсили около 1,0e5 1/Мс, необязательно при измерении с помощью поверхностного плазмонного резонанса, и/или с kd около 1,6e-4 1/с, около 3,3e-5 1/с, около 3,4e-5 1/с, около 3,5e-5 1/с, около 3,6e-5 1/с, около 3,7e-5 1/с, около 3,8e-5 1/с, около 3,9e-5 1/с, около 3,9e-5 1/с, около 4,0e-5 1/с, около 4,1e-5 1/S, около 4,2e-5 1/с, около 4,3e-5 1/с, около 4,4e-5 1/с, около 4,5e-5 1/с, около 4,6e-5 1/с, около 4,7e-5 1/с, около 4,8e-5 1/с, около 4,9e-5 1/с, около 5,0e-5 1/с около 5,1e-5 1/с, около 5,2e-5 1/с, около 5,3e-5 1/с, около 5,4e-5 1/с, около 5,5e-5 1/с, около 5,6e-5 1/с, около 5,7e-5 1/с, около 5,8e-5 1/с, около 5,9e-5 1/с, около 6,0e-5 1/с, около 6,1e-5 1/с, около 6,2e-5 1/с, около 6,3e-5 1/с, около 6,4e-5 1/с или около 6,5e-5 1/с, необязательно, как измерено с помощью поверхностного плазмонного резонанса.
В некоторых вариантах осуществления антитело или антигенсвязывающий фрагмент способны связываться с дегликозилированным S-белком RBD с KD , составляющей около 0,95, около 0,96 нМ, около 0,97 нМ, около 0,98 нМ, около 0,99 нМ, около 1,0 нМ, около 1,1 нМ, около 1,2 нМ, около 1,3 нМ, около 1,4 нМ, около 1,5 нМ или около 1,6 нМ, необязательно, как измерено с помощью поверхностного плазмонного резонанса, и/или с ka, составляющей около (1/с) около 2,5e5, около 2,6e5, около 2,7e5, около 2,8e5, около 2,9e5, около 3,0e5, около 3,1e5, необязательно, как измерено с помощью поверхностного плазмонного резонанса, и/или с kd (1/с) около 2,8e-4, около 2,9e-4, около 3,0e-4, около 3,1e-4, около 3,2e-4, около 3,3e-4, около 3,4e-4, около 3,5e-4, около 3,6e-4, около 3,7e-4, около 3,8e-4, около 3,9e-4, около 4,0e-4, около 4,1e-4, около 4,2e-4, около 4,3e-4, около 4,4e-4, около 4,5e-4, около 4,6e-4 около 4,7e-4, около 4,8e-4, около 4,9e-4 или около 5,0e-4, как необязательно, как измерено с помощью поверхностного плазмонного резонанса.
В некоторых вариантах осуществления для определения связывания с RBD поверхностный плазмонный резонанс включает использование, проводимое с использованием сенсорного чипа с ковалентно иммобилизованным Fc против человека (например, из GE). Буфер может представлять собой 10 мМ HEPES (4-(2-гидроксиэтил)-1-пиперазинэтансульфоновая кислота) с pH 7,4, 150 мМ NaCl, 3 мМ EDTA (этилендиаминтетрауксусная кислота) и 0,05% моющего средства P20. SPR можно проводить при 25 °C. Антитела могут быть разведены от супернатанта жидкости до приблизительно 2 мкг/мл. Концентрации RBD могут составлять 0,8 нМ, 3,1 нМ, 12,5 нМ, 50 нМ и/или 200 нМ.
В некоторых вариантах осуществления антитело или антигенсвязывающий фрагмент способно связываться с рецептор-связывающим доменом (RBD) поверхностного гликопротеина SARS-CoV-2, когда RBD гликозилирован и/или дегликозилирован, где связывание определяли с использованием поверхностного плазмонного резонанса (SPR), где необязательно: (1) SPR выполняли с использованием прибора Biacore T200 с использованием одноциклового кинетического подхода, дополнительно необязательно с 3-минутным периодом инъекции и 20-минутным периодом диссоциации; (2) антитело или его антигенсвязывающий фрагмент захватывали на поверхности; (3) RBD присутствует в концентрации 0,8 нМ, 3,1 нМ, 12,5 нМ., 50 нМ или 200 нМ; (4) антитело или антигенсвязывающий фрагмент связывается с гликозилированным RBD с KD, составляющей около 2,0 нМ, около 1,9 нМ, около 1,8 нМ, около 1,7 нМ, около 1,6 нМ, около 1,5 нМ, около 1,4 нМ, около 1,3 нМ, около 1,2 нМ, около 1,1 нМ, около 1,0 нМ, около 0,9 нМ, около 0,8 нМ, около 0,7 нМ, около 0,6 нМ, около 0,5 нМ или около 0,4 нМ, или с KD, составляющей 0,4±0,05 нМ, или с KD, составляющей 0,45±0,05 нМ, или с KD, составляющей 0,5±0,05 нМ, или с KD 0,6±0,05 нМ, или с KD 0,7±0,05 нМ, или с KD 1,7±0,05 нМ; и/или (5) антитело или антигенсвязывающий фрагмент связывается с дегликозилированной RBD с KD около 37,0 нМ, около 8,0 нМ, около 2,0 нМ, около 1,9 нМ, около 1,8 нМ, около 1,7 нМ, около 1,6 нМ, около 1,5 нМ, около 1,4 нМ, около 1,3 нМ, около 1,2 нМ, около 1,1 нМ, около 1,0 нМ, или около 0,9 нМ, или с KD 37,0±0,05 нМ, или с KD 8,0±0,05 нМ, или с KD 1,0±0,05 нМ, или с KD 0,9±0,05 нМ, или с KD 1,3±0,05 нМ, или с KD 1,8±0,05 нМ, или с KD 1,7±0,05 нМ.
В некоторых вариантах осуществления антитело по настоящему изобретению способно нейтрализовать инфекцию с помощью SARS-CoV-2. В данном контексте "нейтрализующее антитело" представляет собой антитело, которое может нейтрализовать, т.е. предотвращать, ингибировать, уменьшать, задерживать или препятствовать способности патогена инициировать и/или продлять инфекцию в организме хозяина. Нейтрализация может быть количественно оценена, например, путем оценки уровней РНК SARS-CoV-2 в образце (n, например, легкого), оценки вирусной нагрузки SARS-CoV-2 в образце (n, например, легкого), оценки гистопатологии образца (n, например, легкого) или тому подобное. Термины «нейтрализующее антитело» и «антитело, которое нейтрализует» или «антитела, которые нейтрализуют» используются в настоящем документе взаимозаменяемо. В любом из раскрытых в данном документе вариантов осуществления антитело или антигенсвязывающий фрагмент способно предотвращать и/или нейтрализовать инфекцию SARS-CoV-2 на модели инфекции in vitro и/или на животной модели инфекции in vivo (например, с использованием модели сирийского хомячка с интраназальным заражением SARS-CoV-2) и/или у человека. В некоторых вариантах осуществления антитело или антигенсвязывающий фрагмент по настоящему изобретению способно нейтрализовать инфекцию SARS-CoV-2 с IC90 (концентрация, необходимая для ингибирования репликации вируса на 90%), составляющей около 9 мкг/мл. В некоторых вариантах осуществления антитело или антигенсвязывающий фрагмент по настоящему изобретению способно нейтрализовать инфекцию SARS-CoV-2 с IC50, составляющей от около 16 до около 20 мкг/мл. В некоторых вариантах осуществления антитело или антигенсвязывающий фрагмент способно нейтрализовать инфекцию SARS-CoV-2 или вирус, псевдотипированный SARS-CoV-2, с IC50 от около 0,3 до около 0,4 мкг/мл. В некоторых вариантах осуществления антитело или антигенсвязывающий фрагмент, или композиция, содержащая два или более антител или антигенсвязывающих фрагментов по настоящему изобретению, способно нейтрализовать инфекцию SARS-CoV-2 или вирус, псевдотипированный SARS-CoV-2, с IC50 от около 0,07 до около 0,08 мкг/мл.
В некоторых вариантах осуществления антитело или антигенсвязывающий фрагмент (i) распознает эпитоп в мотиве связывания рецептора ACE2 (RBM, SEQ ID NO:167) SARS-CoV-2; (ii) способно блокировать взаимодействие между SARS-CoV-2 и ACE2; (ii) способно связываться с S-белком SARS-CoV-2 с большей авидностью, чем с S-белком коронавируса SARS; (iv) способно окрашивать около 30%, около 35%, около 40%, около 50%, около 55%, около 56%, около 57%, около 58%, около 59%, около 60%, или более клеток-мишеней, экспрессирующих поверхностный гликопротеин SARS-CoV-2, в образце, содержащем около 50 000 клеток-мишеней (например, клетки ExpiCHO) в приблизительно 100 мкл, когда антитело или антигенсвязывающий фрагмент присутствует в концентрации 10 мкг/мл (например, окрашивание, определенное с помощью проточной цитометрии ELISA); (v) распознает эпитоп, который является консервативным в ACE2 RBM SARS-CoV-2 и в ACE2 RBM коронавируса SARS; (vi) является перекрестно-реактивным против коронавируса SARS-CoV-2 и SARS; (vii) распознает эпитоп в поверхностном гликопротеине SARS-CoV-2, который отсутствует в ACE2 RBM; или (viii) любая комбинация (i)-(vii).
В некоторых вариантах осуществления антитело или его антигенсвязывающий фрагмент способно ингибировать взаимодействие между: (i) SARS-CoV-2 и человеческой DC-SIGN; (ii) SARS-CoV-2 и человеческой L-SIGN; (iii) SARS-CoV-2 и человеческим SIGLEC-1; или (iv) любой комбинацией (i)-(iii). Как описано в настоящем документе, DC-SIGN, L-SIGN и SIGLEC-1 могут быть вовлечены в инфекцию SARS-CoV-2 в ролях, включающих роли рецепторов связывания. Ингибирование взаимодействия между SARS-CoV-2 и DC-SIGN, L-SIGN и/или SIGLEC-1 может в некоторых контекстах нейтрализовать инфекцию SARS-CoV-2.
В некоторых вариантах осуществления антитело или его антигенсвязывающий фрагмент способно связываться с поверхностным гликопротеином: (i) SARS-CoV-2 Wuhan-Hu-1 (SEQ ID NO:165); (ii) SARS-CoV-2 B.1.1.7; (iii) SARS-CoV-2 B.1.351; (iv) SARS-CoV-2, содержащего любую одну или более из следующих мутаций по типу замены относительно SEQ ID NO:165: N501Y; S477N; N439K; L452R; E484K; Y453F; A520S; K417N; K417V; S494P; N501T; S477R; V367F; P384L; A522S; A522V; V382L; P330S; T478I; S477I; P479S; или (v) любой комбинации (i)-(iv).
Термины, понятные специалистам в данной области техники, каждый из которых имеет значение, приобретенное в данной области техники, если явно не определено иное в настоящем документе. Например, термин "антитело" относится к интактному антителу, содержащему по меньшей мере две тяжелые (H) цепи и две легкие (L) цепи, соединенные между собой дисульфидными связями, а также любую антигенсвязывающую часть или фрагмент интактного антитела, который обладает или сохраняет способность связываться с молекулой-мишенью антигена, распознаваемой интактным антителом, таким как фрагмент scFv, Fab или Fab'2. Таким образом, термин "антитело" в настоящем документе используется в самом широком смысле и включает поликлональные и моноклональные антитела, включая интактные антитела и их функциональные (антигенсвязывающие) фрагменты антител, включая фрагменты, связывающие фрагменты антигена (Fab), фрагменты F(ab')2, фрагменты Fab', фрагменты Fv, рекомбинантные фрагменты IgG (rIgG), фрагменты одноцепочечных антител, включая одноцепочечные вариабельные фрагменты (scFv), и фрагменты однодоменных антител (sd) (например, sdAb, sdFv, нанотело). Термин охватывает генетически сконструированные и/или иным образом модифицированные формы иммуноглобулинов, такие как интратела, пептидные антитела, химерные антитела, полностью человеческие антитела, гуманизированные антитела и гетероконъюгатные антитела, полиспецифические, например, биспецифические антитела, диатела, триатела, тетратела, тандем ди-scFv и тандем три-scFv. Если не указано иное, термин «антитело» следует понимать как охватывающий его функциональные фрагменты. Термин также охватывает интактные или полноразмерные антитела, включая антитела любого класса или подкласса, включая IgG и его подклассы (IgG1, IgG2, IgG3, IgG4), IgM, IgE, IgA и IgD.
Термины "VL" или "VL" и "VH" или "VH" относятся к вариабельной области связывания из легкой цепи антитела и тяжелой цепи антитела, соответственно. В некоторых вариантах осуществления VL представляет собой класс каппа (κ) (также "VK" в настоящем документе). В некоторых вариантах осуществления VL представляет собой класс лямбда (λ). Вариабельные связывающие области включают дискретные, четко определенные подобласти, известные как "определяющие комплементарность области" (CDR) и "каркасные области" (FR). Термины "область, определяющая комплементарность" и "CDR" являются синонимами "гипервариабельной области" или "HVR" и относятся к последовательностям аминокислот в пределах вариабельных областей антитела, которые, как правило, совместно придают антигенную специфичность и/или аффинность связывания антитела, при этом последовательные CDR (т.е. CDR1 и CDR2, CDR2 и CDR3) отделены друг от друга в первичной структуре каркасной областью. В каждой вариабельной области имеется три CDR (HCDR1, HCDR2, HCDR3; LCDR1, LCDR2, LCDR3; также называемые CDRH и CDRL, соответственно). В некоторых вариантах осуществления антитело VH содержит четыре FR и три CDR следующим образом: FR1-HCDR1-FR2-HCDR2-FR3-HCDR3-FR4; и антитело VL содержит четыре FR и три CDR следующим образом: FR1-LCDR1-FR2-LCDR2-FR3-LCDR3-FR4. Как правило, VH и VL вместе образуют антигенсвязывающий сайт через свои соответствующие CDR.
В контексте настоящего документа "вариант" CDR относится к функциональному варианту последовательности CDR, имеющей до 1-3 аминокислотных замен (например, консервативных или неконсервативных замен), делеций или их комбинаций.
Нумерация CDR и каркасных областей может быть в соответствии с любым известным способом или схемой, такой как схемы нумерации Kabat, Chothia, EU, IMGT и AHo (см., например, Kabat и др., "Sequences of Proteins of Immunological Interest, US Dept. Health and Social Services, Public Health Service National Institutes of Health, 1991,5th ed.; Chothia and Lesk, J. Mol. Biol. 196:901-917 (1987)); Lefranc и др., Dev. Comp. Immunol. 27:55, 2003; Honegger and Plückthun, J. Mol. Bio. 309:657-670 (2001)). Эквивалентные положения остатков можно аннотировать и сравнивать разные молекулы с помощью программного инструмента для нумерации и классификации рецепторов антигена (ANARCI) (2016, Bioinformatics 15:298-300). Соответственно, идентификация CDR примерной последовательности вариабельного домена (VH или VL), предложенной в настоящем документе, в соответствии с одной схемой нумерации не исключает антитело, содержащее CDR того же вариабельного домена, как определено с использованием другой схемы нумерации. В некоторых вариантах осуществления предлагается антитело или антигенсвязывающий фрагмент, которое содержит CDR из последовательности VH согласно любой из SEQ ID NO:113, 1, 9-15, 23, 24, 27, 28-46, 55, 63, 79, 87, 95, 103, 105, 114-120, 129-146, 155, 172, 176-178, 194, 196, 198, 200, 202, 239, и 267, и из последовательности VL согласно любой из SEQ ID NO:168, 5, 47-50, 59, 67, 71-72, 75, 76, 83, 91, 99, 109, 147-150, 159, 182, 190, 234, и 243, как определено с использованием любого известного способа нумерации CDR, включая способы нумерации Kabat, Chothia, EU, IMGT, Martin (улучшенная Chothia), Contact и AHo. В некоторых вариантах осуществления CDR соответствуют способу нумерации IMGT. В некоторых вариантах осуществления CDR соответствуют способу нумерации антител, разработанному Chemical Computing Group (CCG); например, с использованием программного обеспечения Molecular Operating Environment (MOE) (www.chemcomp.com).
В некоторых вариантах осуществления предложено антитело или антигенсвязывающий фрагмент, которые содержат вариабельный домен тяжелой цепи (VH), содержащий CDRH1, CDRH2 и CDRH3, и вариабельный домен легкой цепи (VL), содержащий CDRL1, CDRL2 и CDRL3, где: (i) CDRH1 содержит или состоит из аминокислотной последовательности согласно любой из SEQ ID NO:106, 2, 56, 64, 80, 88, 96, 156, 179, 195 или 240, или ее варианта последовательности, содержащего одну, две или три замены кислоты, одна или более из которых необязательно представляет собой консервативную замену и/или представляет собой замену аминокислоты, кодируемой зародышевой линией; (ii) CDRH2 содержит или состоит из аминокислотной последовательности согласно любой из SEQ ID NO:121, 3, 16-22, 57, 65, 81, 89, 97, 107, 122-126, 157, 180, 197, 199 или 241, или ее варианта последовательности, содержащего одну, две или три аминокислотные замены, одна или более из которых необязательно представляет собой консервативную замену и/или замену аминокислоты, кодируемой зародышевой линией; (iii) CDRH3 содержит или состоит из аминокислотной последовательности согласно любой из SEQ ID NO:108, 4, 25, 26, 58, 66, 82, 90, 98, 104, 127, 128, 158, 181, 201, 203 или 242, или их вариант последовательности, содержащий одну, две или три аминокислотные замены, одна или более из которых необязательно представляет собой консервативную замену и/или является заменой аминокислоты, кодируемой зародышевой линией; (iv) CDRL1 содержит или состоит из аминокислотной последовательности согласно любой из SEQ ID NO:169, 6, 51-54, 60, 68, 73, 74, 84, 92, 100, 110, 160, 183, 235 или 244, или ее варианта последовательности, содержащего одну, две или три аминокислотные замены, одну или более из которых замены необязательно представляют собой консервативную замену и/или являются заменой аминокислоты, кодируемой зародышевой линией; (v) CDRL2 содержит или состоит из аминокислотной последовательности согласно любой из SEQ ID NO:170, 7, 61, 69, 85, 93, 101, 111, 161, 184, 236 или 245, или ее варианта последовательности, содержащего одну, две или три аминокислотные замены, одна или более из которых необязательно представляет собой консервативную замену и/или является заменой аминокислоты, кодируемой зародышевой линией; и/или (vi) CDRL3 содержит или состоит из аминокислотной последовательности согласно любой из SEQ ID NO: №:171, 8, 62, 70, 77, 78, 86, 94, 102, 112, 151, 152, 153, 154, 162, 185, 237 или 246, или их вариант последовательности, содержащий одну, две или три аминокислотные замены, одна или более из которых необязательно представляет собой консервативную замену и/или замену аминокислоты, кодируемой зародышевой линией, где антитело или антигенсвязывающий фрагмент способно связываться с поверхностным гликопротеином SARS-CoV-2, экспрессируемым на поверхности клетки-хозяина, на вирионе или и на том, и другом.
В некоторых вариантах осуществления антитело или антигенсвязывающий фрагмент содержит аминокислотные последовательности VH и VL, которые кодируются:
(i) геном VH1-18 и геном VK3-20, соответственно, или которые кодируются полинуклеотидом, имеющим по меньшей мере 90%, по меньшей мере 91%, по меньшей мере 92%, по меньшей мере 93%, по меньшей мере 94%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98% или по меньшей мере 99% идентичности с VH1-18 и VK3-20, соответственно;
(ii) аллелем VH3-7 и аллелем VL3-25, соответственно, или которые кодируются полинуклеотидом, имеющим по меньшей мере 90%, по меньшей мере 91%, по меньшей мере 92%, по меньшей мере 93%, по меньшей мере 94%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98% или по меньшей мере 99% идентичности с VH3-7 и VL3-25, соответственно;
(iii) аллелем VH3-23 и аллелем VK1-5, соответственно, или которые кодируются полинуклеотидом, имеющим по меньшей мере 90%, по меньшей мере 91%, по меньшей мере 92%, по меньшей мере 93%, по меньшей мере 94%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98% или по меньшей мере 99% идентичности с VH3-23 и VK1-5, соответственно;
(iv) аллелем VH3-13 и аллелем VK1-39, соответственно, или которые кодируются полинуклеотидом, имеющим по меньшей мере 90%, по меньшей мере 91%, по меньшей мере 92%, по меньшей мере 93%, по меньшей мере 94%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98% или по меньшей мере 99% идентичности с VH3-13 и VK1-39, соответственно;
(v) аллелем VH1-18 и аллелем VK3-11, соответственно, или которые кодируются полинуклеотидом, имеющим по меньшей мере 90%, по меньшей мере 91%, по меньшей мере 92%, по меньшей мере 93%, по меньшей мере 94%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98% или по меньшей мере 99% идентичности с VH1-18 и VK3-11, соответственно; или
(vi) аллелем VH1-69 и аллелем VL2-23, соответственно, или которые кодируются полинуклеотидом, имеющим по меньшей мере 90%, по меньшей мере 91%, по меньшей мере 92%, по меньшей мере 93%, по меньшей мере 94%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98% или по меньшей мере 99% идентичности с VH1-69 и VL2-23, соответственно.
В любом из раскрытых в настоящем документе вариантов осуществления антитело или антигенсвязывающий фрагмент способно предотвращать и/или нейтрализовать инфекцию SARS-CoV-2 на модели инфекции in vitro и/или на животной модели инфекции in vivo и/или у человека.
В любом из раскрытых в настоящем документе вариантов осуществления антитело или его антигенсвязывающий фрагмент содержит аминокислотные последовательности CDRH1, CDRH2, CDRH3, CDRL1, CDRL2 и CDRL3 согласно SEQ ID NO: (i) 2-4 и 6-8 или 235-237, соответственно; (ii) 2, любой из 16-22, 4 и 6-8 или 235-237, соответственно; (iii) 2, 3, любой из 25-26 и 6-8 или 235-237, соответственно; (iv) 2-4, 51, 7 и 8, соответственно; (v) 2-4, 52, 7 или 236 и 8 или 237, соответственно; (vi) 2-4, 53, 7 или 236 и 8 или 237, соответственно; (vii) 2-5, 54, 7 или 236 и 8 или 237, соответственно; (viii) 56-58 и 60-62, соответственно; (ix) 64-66 и 68-70, соответственно; (x) 64-66, 73 или 74, 69 и 70, соответственно; (xi) 64-66, 68-69 и 77 или 78, соответственно; (xii) 80-82 и 84-86, соответственно; (xiii) 88-90 и 92-94, соответственно; (xiv) 96-98 и 101-102, соответственно; (xv) 96, 97, 104 и 100-102, соответственно; (xvi) 106-108 и 110-112 или 169-171, соответственно; (xvii) 106, любой из 121-126, 108 и 110-112, соответственно; (xviii) 106, 107, 127 или 128 и 110-112, соответственно; (xix) 106-108, 110, 111 и 151, соответственно; (xx) 106-108, 110, 111 и 152, соответственно; (xxi) 106-108, 110, 111 и 153, соответственно; (xxii) 106-108, 110, 111 и 154., соответственно; (xxiii) 106, 107 или любой из 121-126, 108 или 127 или 128 и 169-171, соответственно; (xxiv) 156-158 и 160-162, соответственно; (xxv) 106, 123, 127 и 169-171, соответственно; (xxvi) 2, 17, 25, 6 или 235 или любой из 51-54, 7 или 236 и 8 или 237, соответственно; (xxvii) 2, 20, 25, 6 или 235 или любой из 51-54, 7 или 236 и 8 или 237, соответственно; (xxviii) 179-181 и 183-185, соответственно, (xxix) 195, 180, 181 и 183-185, соответственно; (xxx) 195, 197, 181 и 183-185, соответственно; (xxxi) 195, 199, 181 и 183-185, соответственно; (xxxii) 195, 197, 201 и 183-185, соответственно; (xxxiii) 195, 197, 203 и 183-185, соответственно; (xxxiv) 195, 199, 201 и 183-185, соответственно; (xxxv) 195, 199, 203 и 183-185, соответственно; (xxxvi) 179, 180, 181 и 183-185, соответственно; (xxxvii) 179, 197, 181 и 183-185, соответственно; (xxxviii) 179, 199, 181 и 183-185, соответственно; (xxxix) 179, 197, 201 и 183-185, соответственно; (xxxx) 179, 197, 203 и 183-185, соответственно; (xxxxi) 179, 199, 201 и 183-185, соответственно; (xxxxii) 179, 199, 203 и 183-185, соответственно; (xxxxiii) 179, 180, 201 и 183-185, соответственно; (xxxxiv) 179, 180, 203 и 183-185, соответственно; и (xxxxv) 240-242 и 244-246, соответственно.
В некоторых вариантах осуществления антитело или антигенсвязывающий фрагмент по настоящему изобретению содержит аминокислотные последовательности CDRH1, CDRH2, CDRH3, CDRL1, CDRL2 и CDRL3, представленные в SEQ ID NO: 80-82 и 84-86, соответственно. В дополнительных вариантах осуществления антитело или антигенсвязывающий фрагмент по настоящему изобретению связывается с S-белком SARS-CoV-2 с KD менее чем около 4,5×10-9 M, менее чем около 5×10-9 M, менее чем около 1×10-10 M, менее чем около 5×10-10 M, менее чем около 1×10-11 M, менее чем около 5×10-11 M, менее чем около 1×10-12 M или менее чем около 5×10-12 M. В дополнительных вариантах осуществления антитело или антигенсвязывающий фрагмент по настоящему изобретению способно нейтрализовать инфекцию SARS-CoV-2 и/или нейтрализовать инфекцию клетки-мишени вирусом, псевдотипированным SARS-CoV-2, с IC50 от около 16 до около 20 мкг/мл.
В некоторых вариантах осуществления антитело или антигенсвязывающий фрагмент согласно настоящему изобретению содержит аминокислотные последовательности CDRH1, CDRH2, CDRH3, CDRL1, CDRL2 и CDRL3, представленные в SEQ ID NO: 106-108 и 169-171 или 106, 121, 108 и 169-171, соответственно. В дополнительных вариантах осуществления антитело или антигенсвязывающий фрагмент по настоящему изобретению связывается с S-белком SARS-CoV-2 с KD менее чем около 4,5×10-9 M, менее чем около 5×10-9 M, менее чем около 1×10-10 M, менее чем около 5×10-10 M, менее чем около 1×10-11 M, менее чем около 5×10-11 M, менее чем около 1×10-12 M или менее чем около 5×10-12 M. В дополнительных вариантах осуществления антитело или антигенсвязывающий фрагмент по настоящему изобретению способно нейтрализовать инфекцию SARS-CoV-2 и/или нейтрализовать инфекцию клетки-мишени вирусом, псевдотипированным SARS-CoV-2, с IC50 от около 0,3 до около 0,4 мкг/мл.
В некоторых вариантах осуществления антитело или его антигенсвязывающий фрагмент по настоящему изобретению содержит CDRH1, CDRH2, CDRH3, CDRL1, CDRL2 и CDRL3, где каждый CDR независимо выбран из соответствующего CDR SARS-CoV-2 S300 mAb, SARS-CoV-2 S300-v1 mAb, SARS-CoV-2 S300-v1.1 mAb, SARS-CoV-2 S300-v1.2 mAb, SARS-CoV-2 S300-v1.3 mAb, SARS-CoV-2 S300-v1.4 mAb, SARS-CoV-2 S300-v1.5 mAb, SARS-CoV-2 S300-v1.6 mAb, SARS-CoV-2 S300-v1.7 mAb, или SARS-CoV-2 S300-v1.8 mAb SARS-CoV-2 S300-v1.9 mAb, SARS-CoV-2 S300-v2 mAb, SARS-CoV-2 S300-v2.1 mAb, SARS-CoV-2 S300-v2.2 mAb, SARS-CoV-2 S300-v2.3 mAb, SARS-CoV-2 S300-v2.4 mAb, SARS-CoV-2 S300-v2.5 mAb, SARS-CoV-2 S300-v2.6 mAb, SARS-CoV-2 S300-v2.7 mAb, SARS-CoV-2 S300-v2.8 mAb, SARS-CoV-2 S300-v2.9 mAb, SARS-CoV-2 S300-v2.10, SARS-CoV-2 S300-v2.11, SARS-CoV-2 S300-v3 mAb , SARS-CoV-2 S300-v3.1 mAb, SARS-CoV-2 S300-v3.2 mAb, SARS-CoV-2 S300-v3.3 mAb, SARS-CoV-2 S300-v3.4 mAb, SARS-CoV-2 S300-v3.5 mAb, SARS-CoV-2 S300-v3.6 mAb, SARS-CoV-2 S300-v3.7 mAb, SARS-CoV-2 S300-v3.8 mAb, SARS-CoV-2 S300-v3.9 mAb, SARS-CoV-2 S300-v10 mAb, SARS-CoV-2 S300-v11 mAb, SARS-CoV-2 S300-v12 mAb, SARS-CoV-2 S300-v13 mAb, SARS-S300-v14 mAb, SARS-CoV-2 S302 mAb, SARS-CoV-2 S303 mAb, SARS-CoV-2 S303-v1 mAb, SARS-CoV-2 S303-v2 mAb, SARS-CoV-2 S303-v3 mAb, SARS-CoV-2 S303-v4 mAb, SARS-CoV-2 S303-v5 mAb, SARS-CoV-2 S304 mAb, SARS-CoV-2 S306 mAb, SARS-CoV-2 S307 mAb, SARS-CoV-2 S308 mAb, SARS-CoV-2 S308-v1 mAb, SARS-CoV-2 S308-v2 mAb, SARS-CoV-2 S309 mAb, SARS-CoV-2 S309-v1 mAb, SARS-CoV-2 S309-v1.1 mAb, SARS-CoV-2 S309-v1.2 mAb, SARS-CoV-2 S309-v1.3 mAb, SARS-CoV-2 S309-v1.4 mAb, SARS-CoV-2 S309-v1.5 mAb, SARS-CoV-2 S309-v1.6 mAb, SARS-CoV-2 S309-v1.7 mAb, SARS-CoV-2 S309-v1.8 mAb, SARS-CoV-2 S309-v2 mAb, SARS-CoV-2 S309-v2.1 mAb, SARS-CoV-2 S309-v2.2 mAb, SARS-CoV-2 S309-v2.3 mAb, SARS-CoV-2 S309-v2.4 mAb, SARS-CoV-2 S309-v2.5 mAb, SARS-CoV-2 S309-v2.6 mAb, SARS-CoV-2 S309-v2.7 mAb, SARS-CoV-2 S309-v2.8 mAb, SARS-CoV-2 309-v2.9 mAb, SARS-CoV-2 S309-v3 mAb, SARS-CoV-2 S309-v3.1 mAb, SARS-CoV-2 S309-v3.2 mAb, SARS-CoV-2 S309-v3.3 mAb, SARS-CoV-2 S309-v3.4 mAb, SARS-CoV-2 S309-v3.5 mAb, SARS-CoV-2 S309-v3.6 mAb, SARS-CoV-2 S309-v3.7 mAb, SARS-CoV-2 S309-v3.8 mAb, SARS-CoV-2 S309-v9 mAb, SARS-CoV-2 S309-v10 mAb, SARS-CoV-2 S309-v11 mAb, SARS-CoV-2 S309-v12 mAb, SARS-CoV-2 S309-v13 mAb, SARS-CoV-2 S310 mAb, SARS-CoV-2 S311 mAb, SARS-CoV-2 S312 mAb, SARS-CoV-2 S315-v1 mAb, SARS-CoV-2 S315-v2 mAb, SARS-CoV-2 S315-v3 mAb, SARS-CoV-2 S315-v4 mAb, SARS-CoV-2 S315-v5 mAb, SARS-CoV-2 S315-v6 mAb, или SARS-CoV-2 S315-v7 mAb, как указано в таблице 2. То есть рассматриваются все комбинации CDR из mAb SARS-CoV-2 и их вариантных последовательностей, представленных в таблице 2.
Примерные антитела по настоящему изобретению включают антитело S309 и его сконструированные варианты. В частных вариантах осуществления антитело или антигенсвязывающий фрагмент содержит CDRH1, CDRH2, CDRH3, CDRL1, CDRL2 и CDRL3, выбранные из любой из аминокислотных последовательностей CDRH1, CDRH2, CDRH3, CDRL1, CDRL2 и CDRL3 (соответственно), представленных в таблице 1.
В некоторых вариантах осуществления антитело или антигенсвязывающий фрагмент содержит: CDRH1, CDRH2 и CDRH3 аминокислотной последовательности VH, представленной в любой из SEQ ID NO:105, 113, 114, 115, 116, 117, 118, 119, 120, 129, 130, 131, 132, 133, 134, 135, 136, 137, 138, 139, 140, 141, 142, 143, 144, 145, 146, 172, и 267; и CDRL1, CDRL2 и CDRL3, представленные в SEQ ID NO:168 (т.е.в соответствии с любым способом нумерации или определения CDR, известным в данной области техники, таким как IMGT, Kabat, Chothia, AHo, North, Contact, CCG, EU или Martin (улучшенная Chothia)).
В дополнительных вариантах осуществления антитело или антигенсвязывающий фрагмент содержит VH, имеющий по меньшей мере 85% идентичности (т.е. 85%, 86, 87, 88, 89, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 или 100%) с аминокислотной последовательностью VH, представленной в таблице 1, и/или VL, имеющий по меньшей мере 85% идентичности (т.е.85%, 86, 87, 88, 89, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 или 100%) с аминокислотной последовательностью VL, представленной в таблице 1. В дополнительных вариантах осуществления антитело или антигенсвязывающий фрагмент содержит VH, имеющий по меньшей мере 90% идентичности с аминокислотной последовательностью VH, представленной в таблице 1, и/или VL, имеющий по меньшей мере 90% идентичности с аминокислотной последовательностью VL, представленной в таблице 1. В дополнительных вариантах осуществления антитело или антигенсвязывающий фрагмент содержит VH, имеющий по меньшей мере 95% идентичности с аминокислотной последовательностью VH, представленной в таблице 1, и/или VL, имеющий по меньшей мере 95% идентичности с аминокислотной последовательностью VL, представленной в таблице 1. В дополнительных вариантах осуществления антитело или антигенсвязывающий фрагмент содержит VH, имеющий по меньшей мере 99% идентичности с аминокислотной последовательностью VH, представленной в таблице 1, и/или VL, имеющий по меньшей мере 99% идентичности с аминокислотной последовательностью VL, представленной в таблице 1. В некоторых вариантах осуществления антитело или антигенсвязывающий фрагмент содержит аминокислотную последовательность VH, выбранную из аминокислотных последовательностей VH, представленных в таблице 1, и аминокислотную последовательность VL, выбранную из аминокислотной последовательности VL, представленной в таблице 1.
ARDYTRGAFFGESLIGGFDN (SEQ ID NO:127);
ARDYTRGAYFGESLIGGFDN (SEQ ID NO:128)
QVQLVQSGAEVKKPGASVKVSCKASGYPFTSYGISWVRQAPGQGLEWMGWISTYQGNTNYAQKFQGRVTMTTDTSTTTGYMELRRLRSDDTAVYYCARDYTRGAWFGESLIGGFDNWGQGTLVTVSS (SEQ ID NO:113)
QVQLVQSGAEVKKPGASVKVSCKASGYPFTSYGISWVRQAPGQGLEWMGWISTYNSNTNYAQKFQGRVTMTTDTSTTTGYMELRRLRSDDTAVYYCARDYTRGAWFGESLIGGFDNWGQGTLVTVSS (SEQ ID NO:114)
QVQLVQSGAEVKKPGASVKVSCKASGYPFTSYGISWVRQAPGQGLEWMGWISTYNANTNYAQKFQGRVTMTTDTSTTTGYMELRRLRSDDTAVYYCARDYTRGAWFGESLIGGFDNWGQGTLVTVSS (SEQ ID NO:115)
QVQLVQSGAEVKKPGASVKVSCKASGYPFTSYGISWVRQAPGQGLEWMGWISTYNQNTNYAQKFQGRVTMTTDTSTTTGYMELRRLRSDDTAVYYCARDYTRGAWFGESLIGGFDNWGQGTLVTVSS (SEQ ID NO:116)
QVQLVQSGAEVKKPGASVKVSCKASGYPFTSYGISWVRQAPGQGLEWMGWISTYLGNTNYAQKFQGRVTMTTDTSTTTGYMELRRLRSDDTAVYYCARDYTRGAWFGESLIGGFDNWGQGTLVTVSS (SEQ ID NO:117)
QVQLVQSGAEVKKPGASVKVSCKASGYPFTSYGISWVRQAPGQGLEWMGWISTYTGNTNYAQKFQGRVTMTTDTSTTTGYMELRRLRSDDTAVYYCARDYTRGAWFGESLIGGFDNWGQGTLVTVSS (SEQ ID NO:118)
QVQLVQSGAEVKKPGASVKVSCKASGYPFTSYGISWVRQAPGQGLEWMGWISTYNGNTNYAQKFQGRVTMTTDTSTTTGYMELRRLRSDDTAVYYCARDYTRGAFFGESLIGGFDNWGQGTLVTVSS (SEQ ID NO:119)
QVQLVQSGAEVKKPGASVKVSCKASGYPFTSYGISWVRQAPGQGLEWMGWISTYNGNTNYAQKFQGRVTMTTDTSTTTGYMELRRLRSDDTAVYYCARDYTRGAYFGESLIGGFDNWGQGTLVTVSS (SEQ ID NO:120)
QVQLVQSGAEVKKPGASVKVSCKASGYPFTSYGISWVRQAPGQGLEWMGFISTYNGNTNYAQKFQGRVTMTTDTSTTTGYMELRRLRSDDTAVYYCARDYTRGAWFGESLIGGFDNWGQGTLVTVSS (SEQ ID NO:129)
QVQLVQSGAEVKKPGASVKVSCKASGYPFTSYGISWVRQAPGQGLEWMGFISTYQGNTNYAQKFQGRVTMTTDTSTTTGYMELRRLRSDDTAVYYCARDYTRGAWFGESLIGGFDNWGQGTLVTVSS (SEQ ID NO:130)
QVQLVQSGAEVKKPGASVKVSCKASGYPFTSYGISWVRQAPGQGLEWMGFISTYNSNTNYAQKFQGRVTMTTDTSTTTGYMELRRLRSDDTAVYYCARDYTRGAWFGESLIGGFDNWGQGTLVTVSS (SEQ ID NO:131)
QVQLVQSGAEVKKPGASVKVSCKASGYPFTSYGISWVRQAPGQGLEWMGFISTYNANTNYAQKFQGRVTMTTDTSTTTGYMELRRLRSDDTAVYYCARDYTRGAWFGESLIGGFDNWGQGTLVTVSS (SEQ ID NO:132)
QVQLVQSGAEVKKPGASVKVSCKASGYPFTSYGISWVRQAPGQGLEWMGFISTYNQNTNYAQKFQGRVTMTTDTSTTTGYMELRRLRSDDTAVYYCARDYTRGAWFGESLIGGFDNWGQGTLVTVSS (SEQ ID NO:133)
QVQLVQSGAEVKKPGASVKVSCKASGYPFTSYGISWVRQAPGQGLEWMGFISTYLGNTNYAQKFQGRVTMTTDTSTTTGYMELRRLRSDDTAVYYCARDYTRGAWFGESLIGGFDNWGQGTLVTVSS (SEQ ID NO:134)
QVQLVQSGAEVKKPGASVKVSCKASGYPFTSYGISWVRQAPGQGLEWMGFISTYTGNTNYAQKFQGRVTMTTDTSTTTGYMELRRLRSDDTAVYYCARDYTRGAWFGESLIGGFDNWGQGTLVTVSS (SEQ ID NO:135)
QVQLVQSGAEVKKPGASVKVSCKASGYPFTSYGISWVRQAPGQGLEWMGFISTYNGNTNYAQKFQGRVTMTTDTSTTTGYMELRRLRSDDTAVYYCARDYTRGAFFGESLIGGFDNWGQGTLVTVSS (SEQ ID NO:136)
QVQLVQSGAEVKKPGASVKVSCKASGYPFTSYGISWVRQAPGQGLEWMGFISTYNGNTNYAQKFQGRVTMTTDTSTTTGYMELRRLRSDDTAVYYCARDYTRGAYFGESLIGGFDNWGQGTLVTVSS (SEQ ID NO:137)
QVQLVQSGAEVKKPGASVKVSCKASGYPFTSYGISWVRQAPGQGLEWMGYISTYNGNTNYAQKFQGRVTMTTDTSTTTGYMELRRLRSDDTAVYYCARDYTRGAWFGESLIGGFDNWGQGTLVTVSS (SEQ ID NO:138)
QVQLVQSGAEVKKPGASVKVSCKASGYPFTSYGISWVRQAPGQGLEWMGYISTYQGNTNYAQKFQGRVTMTTDTSTTTGYMELRRLRSDDTAVYYCARDYTRGAWFGESLIGGFDNWGQGTLVTVSS (SEQ ID NO:139)
QVQLVQSGAEVKKPGASVKVSCKASGYPFTSYGISWVRQAPGQGLEWMGYISTYNSNTNYAQKFQGRVTMTTDTSTTTGYMELRRLRSDDTAVYYCARDYTRGAWFGESLIGGFDNWGQGTLVTVSS (SEQ ID NO:140)
QVQLVQSGAEVKKPGASVKVSCKASGYPFTSYGISWVRQAPGQGLEWMGYISTYNANTNYAQKFQGRVTMTTDTSTTTGYMELRRLRSDDTAVYYCARDYTRGAWFGESLIGGFDNWGQGTLVTVSS (SEQ ID NO:141)
QVQLVQSGAEVKKPGASVKVSCKASGYPFTSYGISWVRQAPGQGLEWMGYISTYNQNTNYAQKFQGRVTMTTDTSTTTGYMELRRLRSDDTAVYYCARDYTRGAWFGESLIGGFDNWGQGTLVTVSS (SEQ ID NO:142)
QVQLVQSGAEVKKPGASVKVSCKASGYPFTSYGISWVRQAPGQGLEWMGYISTYLGNTNYAQKFQGRVTMTTDTSTTTGYMELRRLRSDDTAVYYCARDYTRGAWFGESLIGGFDNWGQGTLVTVSS (SEQ ID NO:143)
QVQLVQSGAEVKKPGASVKVSCKASGYPFTSYGISWVRQAPGQGLEWMGYISTYTGNTNYAQKFQGRVTMTTDTSTTTGYMELRRLRSDDTAVYYCARDYTRGAWFGESLIGGFDNWGQGTLVTVSS (SEQ ID NO:144)
QVQLVQSGAEVKKPGASVKVSCKASGYPFTSYGISWVRQAPGQGLEWMGYISTYNGNTNYAQKFQGRVTMTTDTSTTTGYMELRRLRSDDTAVYYCARDYTRGAFFGESLIGGFDNWGQGTLVTVSS (SEQ ID NO:145)
QVQLVQSGAEVKKPGASVKVSCKASGYPFTSYGISWVRQAPGQGLEWMGYISTYNGNTNYAQKFQGRVTMTTDTSTTTGYMELRRLRSDDTAVYYCARDYTRGAYFGESLIGGFDNWGQGTLVTVSS (SEQ ID NO:146)
QVQLVQSGAEVKKPGASVKVSCKASGYPFTSYGISWVRQAPGQGLEWMGFISTYNANTNYAQKFQGRVTMTTDTSTTTGYMELRRLRSDDTAVYYCARDYTRGAFFGESLIGGFDNWGQGTLVTVSS (SEQ ID NO:172)
QVQLVQSGAEVKKPGASVKVSCKASGYPFTSYGISWVRQAPGQGLEWMGX1ISTYX2X3NTNYAQKFQGRVTMTTDTSTTTGYMELRRLRSDDTAVYYCARDYTRGAX4FGESLIGGFDNWGQGTLVTVSS
Где X1 представляет собой W, F или Y; X2 представляет собой N, Q, L или T; X3 представляет собой G, S, A или Q; X4 представляет собой W, F или Y
В частных вариантах осуществления антитело или антигенсвязывающий фрагмент содержит CDRH1, CDRH2 и CDRH3 согласно SEQ ID NO:106, 107 или 121 или 122 или 123 или 124 или 125 или 126, и 108 или 127 или 128, соответственно, и CDRL1, CDRL2 и CDRL3 согласно SEQ ID NO:169-171, соответственно. В некоторых вариантах осуществления указанное антитело или антигенсвязывающий фрагмент содержит аминокислотные последовательности CDRH1, CDRH2, CDRH3, CDRL1, CDRL2 и CDRL3, представленные в: (a) SEQ ID NO:106, 121, 108, 169, 170 и 171, соответственно; (b) SEQ ID NO: 106, 121, 127, 169, 170 и 171, соответственно; (c) SEQ ID NO: 106, 121, 128, 169, 170 и 171, соответственно;
(d) SEQ ID NO: 106, 107, 108, 169, 170 и 171, соответственно; (e) SEQ ID NO: 106, 107, 127, 169, 170 и 171, соответственно; (f) SEQ ID NO: 106, 107, 128, 169, 170 и 171, соответственно; (g) SEQ ID NO: 106, 122, 108, 169, 170 и 171, соответственно; (h) SEQ ID NO: 106, 122, 127, 169, 170 и 171, соответственно;
(i) SEQ ID NO: 106, 122, 128, 169, 170 и 171, соответственно; (j) SEQ ID NO: 106, 123, 108, 169, 170 и 171, соответственно; (k) SEQ ID NO: 106, 123, 127, 169, 170 и 171, соответственно; (l) SEQ ID NO: 106, 123, 128, 169, 170 и 171, соответственно;
(m) SEQ ID NO: 106, 124, 108, 169, 170 и 171, соответственно; (n) SEQ ID NO: 106, 124, 127, 169, 170 и 171, соответственно; (o) SEQ ID NO: 106, 124, 128, 169, 170 и 171, соответственно; (p) SEQ ID NO: 106, 125, 108, 169, 170 и 171, соответственно; (q) SEQ ID NO: 106, 125, 127, 169, 170 и 171, соответственно;
(r) SEQ ID NO: 106, 125, 128, 169, 170 и 171, соответственно; (s) SEQ ID NO: 106, 126, 108, 169, 170 и 171, соответственно; (t) SEQ ID NO: 106, 126, 127, 169, 170 и 171, соответственно; или (u) SEQ ID NO: 106, 126, 128, 169, 170 и 171, соответственно.
В дополнительных вариантах осуществления VH содержит или состоит из аминокислотной последовательности, представленной в любой из SEQ ID NO:105, 113, 114, 115, 116, 117, 118, 119, 120, 129, 130, 131, 132, 133, 134, 135, 136, 137, 138, 139, 140, 141, 142, 143, 144, 145, 146, 172, и 267, и VL содержит или состоит из аминокислотной последовательности, представленной в SEQ ID NO:168.
Термин "CL" относится к "константной области легкой цепи иммуноглобулина" или "константной области легкой цепи", т.е. константной области из легкой цепи антитела. Термин "CH" относится к "константной области тяжелой цепи иммуноглобулина" или "константной области тяжелой цепи", которая дополнительно делится в зависимости от изотипа антитела на CH1, CH2 и CH3 (IgA, IgD, IgG) или домены CH1, CH2, CH3 и CH4 (IgE, IgM). Fc-область тяжелой цепи антитела описана далее в настоящем документе. В любом из раскрытых в настоящем документе вариантов осуществления антитело или антигенсвязывающий фрагмент согласно настоящему изобретению содержит любую одну или более из CL, CH1, CH2 и CH3. В некоторых вариантах осуществления CL содержит аминокислотную последовательность, имеющую 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентичности с аминокислотной последовательностью SEQ ID NO:174 или SEQ ID NO:193. В некоторых вариантах осуществления CH1-CH2-CH3 содержит аминокислотную последовательность, имеющую 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентичности с аминокислотной последовательностью SEQ ID NO:173 или SEQ ID NO:175 или SEQ ID NO:265 или SEQ ID NO:266. Следует понимать, что, например, продукция в линии клеток млекопитающих может удалять один или более С-концевой лизин тяжелой цепи антитела (см., например, Liu и др. mAb 6(5): 1145-1154(2014)). Соответственно, антитело или антигенсвязывающий фрагмент по настоящему изобретению может содержать тяжелую цепь, CH1-CH3, CH3 или полипептид Fc, где С-концевой остаток лизина присутствует или отсутствует; другими словами, они охватывают варианты осуществления, в которых С-концевой остаток тяжелой цепи, CH1-CH3 или полипептид Fc не является лизином, и варианты осуществления, где лизин представляет собой С-концевой остаток. В некоторых вариантах осуществления композиция содержит множество антител и/или антигенсвязывающих фрагментов по настоящему изобретению, где одно или более антител или антигенсвязывающих фрагментов не содержит остаток лизина на С-конце тяжелой цепи, CH1-CH3 или полипептида Fc, и где одно или более антител или антигенсвязывающих фрагментов содержит остаток лизина на С-конце тяжелой цепи, CH1-CH3 или полипептида Fc.
"Fab" (антигенсвязывающий фрагмент) представляет собой часть антитела, которая связывается с антигенами и включает вариабельную область и CH1 тяжелой цепи, связанной с легкой цепью посредством межцепочечной дисульфидной связи. Каждый фрагмент Fab является одновалентным по отношению к связыванию антигена, то естьон имеет один антигенсвязывающий сайт. Лечение пепсином антитела дает один большой фрагмент F(ab')2, который примерно соответствует двум дисульфид-связанным Fab-фрагментам, обладающим двухвалентной антигенсвязывающей активностью и все еще способным к перекрестному связыванию антигена. Как Fab, так и F(ab’)2 являются примерами «антигенсвязывающих фрагментов». Фрагменты Fab' отличаются от фрагментов Fab наличием дополнительных нескольких остатков на карбоксильном конце домена CH1, включая один или более цистеинов из шарнирной области антитела. Fab'-SH в настоящем документе обозначает Fab', в котором остаток (остатки) цистеина константных доменов несут свободную тиольную группу. F(ab')2-фрагменты антител первоначально были получены в виде пар Fab'-фрагментов, имеющих между собой шарнирные остатки цистеина. Известны также другие химические сочетания фрагментов антител.
Фрагменты Fab могут быть соединены, например, с помощью пептидного линкера, с образованием одноцепочечного Fab, также называемого в настоящем документе "scFab". В этих вариантах осуществления межцепочечная дисульфидная связь, которая присутствует в нативном Fab, может отсутствовать, и линкер полностью или частично служит для связывания или соединения фрагментов Fab в одной полипептидной цепи. Fab-фрагмент, полученный из тяжелой цепи (например, содержащий, состоящий из или состоящий по существу из VH + CH1 или "Fd") и Fab-фрагмент, полученный из легкой цепи (например, содержащий, состоящий из или состоящий по существу из VL + CL), могут быть связаны в любом расположении с образованием scFab. Например, scFab может быть расположен в направлении от N-конца к C-концу в соответствии с (Fab-фрагмент тяжелой цепи - линкер - Fab-фрагмент легкой цепи) или (Fab-фрагмент легкой цепи - линкер - Fab-фрагмент тяжелой цепи). Пептидные линкеры и примерные линкерные последовательности для применения в scFab более подробно обсуждаются в настоящем документе.
ScFab может содержать любую комбинацию последовательностей VH и VL или любую комбинацию последовательностей CDRH1, CDRH2, CDRH3, CDRL1, CDRL2 и CDRL3, описанных в настоящем документе. В некоторых вариантах осуществления scFab содержит последовательность VH, представленную в SEQ ID NO: 105 или SEQ ID NO: 113, и последовательность VL, представленную в SEQ ID NO: 168. В некоторых вариантах осуществления изобретения scFab содержит последовательность CDRH1, представленную в SEQ ID NO: 106, последовательность CDRH2, представленную в SEQ ID NO: 107 или 121, последовательность CDRH3, представленную в SEQ ID NO: 108, последовательность CDRL1, представленную в SEQ ID NO: 169, последовательность CDRL2, представленную в SEQ ID NO: 170, и последовательность CDRL3, представленную в SEQ ID NO: 171. В некоторых вариантах осуществления scFab содержит аминокислотную последовательность, имеющую по меньшей мере 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентичности с аминокислотной последовательностью, представленной в любой из SEQ ID NO: 218-219 или 226-227.
"Fv" представляет собой небольшой фрагмент антитела, который содержит полный антиген-распознающий и антиген-связывающий сайт. Этот фрагмент обычно состоит из димера одного домена вариабельной области тяжелой и одной легкой цепи в тесной нековалентной ассоциации. Однако даже один вариабельный домен (или половина Fv, содержащая только три CDR, специфичных к антигену) обладает способностью распознавать и связывать антиген, хотя обычно с более низкой аффинностью, чем целый сайт связывания.
"Одноцепочечный Fv", также сокращенно "sFv" или "scFv", представляют собой фрагменты антител, которые содержат домены антител VH и VL, соединенные в одну полипептидную цепь. В некоторых вариантах осуществления полипептид scFv содержит полипептидный линкер, расположенный между доменами VH и VL и связывающий их, что позволяет scFv сохранять или формировать желаемую структуру для связывания антигена. Такой пептидный линкер может быть включен в слитый полипептид с использованием стандартных методик, хорошо известных в данной области техники. Дополнительно или поочередно, Fv может иметь дисульфидную связь, образованную между VH и VL и стабилизирующим их. Обзор scFv см. в Pluckthun in The Pharmacology of Monoclonal Antibodies, vol. 113, Rosenburg and Moore eds., Springer-Verlag, New York, pp. 269-315 (1994); Borrebaeck 1995, infra. В некоторых вариантах осуществления указанное антитело или его антигенсвязывающий фрагмент содержит scFv, содержащий домен VH, домен VL и пептидный линкер, связывающий домен VH с доменом VL. В частных вариантах осуществления scFv содержит домен VH, связанный с доменом VL пептидным линкером, который может находиться в ориентации VH-линкер-VL или в ориентации VL-линкер-VH. Любой scFv по настоящему изобретению может быть сконструирован таким образом, что С-концевой домен VL связан короткой пептидной последовательностью с N-концом домена VH или наоборот (т.е., (N)VL(C)-линкер-(N)VH(C) или (N)VH(C)-линкер-(N)VL(C). В качестве альтернативы, в некоторых вариантах осуществления линкер может быть связан с N-концевой частью или концом домена VH, домена VL или обоими.
Последовательности пептидных линкеров могут быть выбраны, например, на основании: (1) их способности принимать гибкую расширенную конформацию; (2) их неспособности или отсутствия способности принимать вторичную структуру, которая может взаимодействовать с функциональными эпитопами на первом и втором полипептидах и/или на молекуле-мишени; и/или (3) отсутствия или относительного отсутствия гидрофобных или заряженных остатков, которые могут взаимодействовать с полипептидами и/или молекулой-мишенью. Другие соображения, касающиеся конструкции линкера (например, длина), могут включать конформацию или диапазон конформаций, в которых VH и VL могут образовывать функциональный антигенсвязывающий сайт. В некоторых вариантах осуществления пептидные линкерные последовательности содержат, например, остатки Gly, Asn и Ser. Другие близкие нейтральные аминокислоты, такие как Thr и Ala, также могут применяться в линкерной последовательности. Другие аминокислотные последовательности, которые могут быть использованы в качестве линкера, включают те, которые описаны в Maratea и др., Gene 40:39 46 (1985); Murphy м др., Proc. Natl. Acad. Sci. USA 83:8258 8262 (1986); патент США № 4935233 и патенте США No. 4,751,180. Другие иллюстративные и неограничивающие примеры линкеров могут включать, например, Glu-Gly-Lys-Ser-Ser-Gly-Ser-Gly-Ser-Glu-Ser-Lys-Val-Asp (SEQ ID NO: 215) (Chaudhary и др., Proc. Natl. Acad. Sci. USA 87:1066-1070 (1990)) и Lys-Glu-Ser-Gly-Ser-Val-Ser-Ser-Glu-Gln-Leu-Ala-Gln-Phe-Arg-Ser-Leu-Asp (SEQ ID NO: 216) (Bird и др., Science 242:423-426 (1988)) и пентамер Gly-Gly-Gly-Gly-Ser (SEQ ID NO: 217), когда они присутствуют в одной итерации или повторяются от 1 до 5 или более раз или более; см., например, SEQ ID NO: 213. Может быть использован любой подходящий линкер, и, как правило, он может иметь длину около 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 15 23, 24, 25, 26, 27, 28, 29, 30, 40, 50, 60, 70, 80, 90, 100 аминокислот или менее чем около 200 аминокислот в длину и предпочтительно будет содержать гибкую структуру (может обеспечивать гибкость и пространство для конформационного движения между двумя областями, доменами, мотивами, фрагментами или модулями, соединенными линкером) и предпочтительно будет биологически инертным и/или иметь низкий риск иммуногенности у человека. Иллюстративные линкеры включают те, которые содержат или состоят из аминокислотной последовательности, изложенной в любой одной или более из SEQ ID NO: 206-217. В некоторых вариантах осуществления изобретения линкер содержит или состоит из аминокислотной последовательности, имеющей по меньшей мере 75% (т.е. по меньшей мере около 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более) идентичности с аминокислотной последовательностью, представленной в любой из SEQ ID NO: 206-217.
scFv может быть сконструирован с использованием любой комбинации последовательностей VH и VL или любой комбинации последовательностей CDRH1, CDRH2, CDRH3, CDRL1, CDRL2 и CDRL3, описанных в настоящем документе. В некоторых вариантах осуществления scFv содержит последовательность VH, представленную в SEQ ID NO: 105 или SEQ ID NO: 113, и последовательность VL, представленную в SEQ ID NO: 168. В некоторых вариантах осуществления изобретения scFab содержит последовательность CDRH1, представленную в SEQ ID NO: 106, последовательность CDRH2, представленную в SEQ ID NO: 107 или 121, последовательность CDRH3, представленную в SEQ ID NO: 108, последовательность CDRL1, представленную в SEQ ID NO: 169, последовательность CDRL2, представленную в SEQ ID NO: 170, и последовательность CDRL3, представленную в SEQ ID NO: 171. В некоторых вариантах осуществления scFv может содержать аминокислотную последовательность, представленную в SEQ ID NO: 220-221 или SEQ ID NO: 228-229.
В некоторых вариантах осуществления изобретения линкерные последовательности не требуются; например, когда первый и второй полипептиды имеют несущественные N-концевые аминокислотные области, которые могут быть использованы для разделения функциональных доменов и предотвращения стерической интерференции.
Во время разработки антитела, ДНК в в локусах гена изменения (V), соединения (J), и разнообразия (D) может быть перестроена, и могут происходить вставки и/или делеции нуклеотидов в кодирующей последовательности. Соматические мутации могут быть кодированы полученной последовательностью и могут быть идентифицированы со ссылкой на соответствующую известную последовательность зародышевой линии. В некоторых контекстах соматические мутации, которые не являются критическими для необходимого свойства антитела (например, связывание с антигеном SARS-CoV-2) или которые придают нежелательное свойство антителу (например, повышенный риск иммуногенности у субъекта, которому вводят антитело), или и то, и другое, могут быть заменены соответствующей аминокислотой, кодируемой зародышевой линией, или другой аминокислотой, так что необходимое свойство антитела улучшается или поддерживается, а нежелательное свойство антитела уменьшается или нейтрализуется. Таким образом, в некоторых вариантах осуществления антитело или антигенсвязывающий фрагмент по настоящему изобретению содержит по меньшей мере еще одну аминокислоту, кодируемую зародышевой линией в вариабельной области по сравнению с исходным антителом или антигенсвязывающим фрагментом, при условии, что исходное антитело или антигенсвязывающий фрагмент содержит одну или более соматических мутаций. Аминокислотные последовательности вариабельной области и CDR примерных антител против SARS-CoV-2 по настоящему изобретению представлены в таблице 2 настоящего документа.
В некоторых вариантах осуществления антитело или антигенсвязывающий фрагмент содержит аминокислотную модификацию (например, мутацию по типу замены) для устранения нежелательного риска окисления, дезамидирования и/или изомеризации.
В настоящем документе также предложены варианты антител, которые содержат одно или более аминокислотных изменений в вариабельной области (например, VH, VL, каркасной области или CDR) по сравнению с раскрытым в настоящем документе ("исходным") антителом, где вариантное антитело способно связываться с антигеном SARS-CoV-2.
В некоторых вариантах осуществления изобретения VH содержит или состоит из аминокислотной последовательности, имеющей по меньшей мере 85% (т.е.85%, 86, 87, 88, 89, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 или 100%) идентичности с аминокислотной последовательностью согласно любой из SEQ ID NO: 1, 9-15, 23, 24, 27, 28-46, 55, 63, 79, 87, 95, 103, 105, 113-120, 129-146, 155, 172, 176-178, 194, 196, 198, 200, 202 и 239, где вариация необязательно ограничена одной или более каркасными областями и/или вариация содержит одну или более замен аминокислоты, кодируемой зародышевой линией; и/или (ii) VL содержит или состоит из аминокислотной последовательности, имеющей по меньшей мере 85% (т.е.85%, 86, 87, 88, 89, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 или 100%) идентичности с аминокислотной последовательностью согласно любой из SEQ ID NO: 5, 47-50, 59, 67, 71-72, 75, 76, 83, 91, 99, 109, 147-150, 159, 168, 182, 190, 234 и 243, где вариация необязательно ограничивается одной или более каркасными областями и/или вариация содержит одну или более замену аминокислоты, кодируемой зародышевой линией.
В некоторых вариантах осуществления изобретения антитело или антигенсвязывающий фрагмент содержит аминокислотные последовательности VH и VL, которые кодируются или являются такими же, как аминокислотные последовательности VH и VL, которые кодируются:
(i) геном VH1-18 и геном VK3-20, соответственно, или которые кодируются полинуклеотидом, имеющим по меньшей мере 90%, по меньшей мере 91%, по меньшей мере 92%, по меньшей мере 93%, по меньшей мере 94%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98% или по меньшей мере 99% идентичности с VH1-18 и VK3-20, соответственно;
(ii) аллелем VH3-7 и аллелем VL3-25, соответственно, или которые кодируются полинуклеотидом, имеющим по меньшей мере 90%, по меньшей мере 91%, по меньшей мере 92%, по меньшей мере 93%, по меньшей мере 94%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98% или по меньшей мере 99% идентичности с VH3-7 и VL3-25, соответственно;
(iii) аллелем VH3-23 и аллелем VK1-5, соответственно, или которые кодируются полинуклеотидом, имеющим по меньшей мере 90%, по меньшей мере 91%, по меньшей мере 92%, по меньшей мере 93%, по меньшей мере 94%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98% или по меньшей мере 99% идентичности с VH3-23 и VK1-5, соответственно;
(iv) аллелем VH3-13 и аллелем VK1-39, соответственно, или которые кодируются полинуклеотидом, имеющим по меньшей мере 90%, по меньшей мере 91%, по меньшей мере 92%, по меньшей мере 93%, по меньшей мере 94%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98% или по меньшей мере 99% идентичности с VH3-13 и VK1-39, соответственно;
(v) аллелем VH1-18 и аллелем VK3-11, соответственно, или которые кодируются полинуклеотидом, имеющим по меньшей мере 90%, по меньшей мере 91%, по меньшей мере 92%, по меньшей мере 93%, по меньшей мере 94%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98% или по меньшей мере 99% идентичности с VH1-18 и VK3-11, соответственно; или
(vi) аллелем VH1-69 и аллелем VL2-23, соответственно, или которые кодируются полинуклеотидом, имеющим по меньшей мере 90%, по меньшей мере 91%, по меньшей мере 92%, по меньшей мере 93%, по меньшей мере 94%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98% или по меньшей мере 99% идентичности с VH1-69 и VL2-23, соответственно.
В некоторых вариантах осуществления VH содержит или состоит из любой аминокислотной последовательности VH, представленной в таблице 2, и VL содержит или состоит из любой аминокислотной последовательности VL, приведенной в таблице 2. В частных вариантах осуществления VH и VL содержат или состоят из аминокислотных последовательностей согласно SEQ ID NO: (i) 1 и 5 или 234, соответственно; (ii) любой из 9-15 и 5 или 234, соответственно; (iii) 23 или 24 и 5 или 234, соответственно; (iv) 27 и 5 или 234, соответственно; (v) любой из 28-46 и 5 или 234, соответственно; (vi) 1 и любой из 47-50, соответственно; (vii) любой из 9-15 и любой из 47-50, соответственно; (viii) 23 или 24 и любой из 47-50, соответственно; (ix) 27 и любой из 47-50, соответственно; (x) любой из 28-46 и любой из 47-50, соответственно; (xi) 55 и 59, соответственно; (xii) 63 и 67, соответственно; (xiii) 63 и 71 или 72, соответственно; (xiv) 63 и 75 или 76, соответственно; (xv) 79 и 83, соответственно; (xvi) 87 и 91, соответственно; (xvii) 95 и 99, соответственно; (xviii) 103 и 99, соответственно; (xiv) 105 и 109 или 168, соответственно; (xx) любой из 113-120 и 109 или 168, соответственно; (xxi) 129 и 109 или 168, соответственно; (xxii) любой из 130-146 и 109 или 168, соответственно; (xxiii) 105 и любой из 147-150, соответственно; (xxiv) любой 113-120 и любой из 147-150, соответственно; (xxv) любой из 130-146 и любой из 147-150, соответственно; (xxvi) 155 и 159, соответственно; (xxvii) 172 и 168, соответственно; (xxviii) 176 или 177 и 5 или любой из 47-50, соответственно; (xxix) 178 и 182 или 190, соответственно (т.е. 178 и 182, соответственно, или 178 и 190, соответственно); (xxx) 194 и 182, соответственно; (xxxi) 196 и 182, соответственно; (xxxii) 198 и 182, соответственно; (xxxiii) 200 и 182, соответственно; (xxxiv) 202 и 182, соответственно; или (xxxv) 239 и 243, соответственно.
В некоторых вариантах осуществления антиген или антигенсвязывающий фрагмент согласно настоящему изобретению содержит VH, содержащий или состоящий из аминокислотной последовательности согласно SEQ ID NO:79, и VL, содержащий или состоящую из аминокислотной последовательности согласно SEQ ID NO:83. В дополнительных вариантах осуществления антиген или антигенсвязывающий фрагмент по настоящему изобретению содержит VH, содержащий или состоящий из аминокислотной последовательности согласно SEQ ID NO:79, и VL, содержащий или состоящий из аминокислотной последовательности согласно SEQ ID NO:83, и связывается с S-белком SARS-CoV-2 с KD менее чем около 4,5×10-9 M, менее чем около 5×10-9 M, менее чем около 1×10-10 M, менее чем около 5×10-10 M, менее чем около 1×10-11 M, менее чем около 5×10-11 M, менее чем около 1×10-12 M или менее чем около 5×10-12 M. В дополнительных вариантах осуществления антиген или антигенсвязывающий фрагмент по настоящему изобретению содержит VH, содержащий или состоящий из аминокислотной последовательности согласно SEQ ID NO: 79, и VL, содержащий или состоящий из аминокислотной последовательности согласно SEQ ID NO: 83 и способную нейтрализовать инфекцию SARS-CoV-2 или вирус, псевдотипированный SARS-CoV-2, с IC50 от около 16 до около 20 мкг/мл.
В некоторых вариантах осуществления антитело или антигенсвязывающий фрагмент по настоящему изобретению содержит VH, содержащий или состоящий из аминокислотной последовательности согласно SEQ ID NO:105, и VL, содержащий или состоящий из аминокислотной последовательности согласно SEQ ID NO:168. В дополнительных вариантах осуществления антитело или антигенсвязывающий фрагмент по настоящему изобретению содержит VH, содержащий или состоящий из аминокислотной последовательности согласно SEQ ID NO:105, и VL, содержащий или состоящий из аминокислотной последовательности согласно SEQ ID NO:168, и связывается с S-белком SARS-CoV-2 с KD менее чем около 4,5×10-9 M, менее чем около 5×10-9 M, менее чем около 1×10-10 M, менее чем около 5×10-10 M, менее чем около 1×10-11 M, менее чем около 5×10-11 M, менее чем около 1×10-12 M или менее чем около 5×10-12 M. В дополнительных вариантах осуществления антитело или антигенсвязывающий фрагмент по настоящему изобретению содержит VH, содержащий или состоящий из аминокислотной последовательности согласно SEQ ID NO:105, и VL, содержащий или состоящий из аминокислотной последовательности согласно SEQ ID NO:168 и способную нейтрализовать инфекцию SARS-CoV-2 или вирус, псевдотипированный SARS-CoV-2, с IC50 от около 0,3 до около 0,4 мкг/мл.
В некоторых вариантах осуществления антитело или антигенсвязывающий фрагмент по настоящему изобретению содержит VH, содержащий или состоящий из аминокислотной последовательности согласно SEQ ID NO:105, и VL, содержащий или состоящий из аминокислотной последовательности согласно SEQ ID NO:168, и связывается с S-белком RBD SARS-CoV-2 с EC50 (полумаксимальная эффективная концентрация) от около 11 до около 25 нг/мл. В некоторых вариантах осуществления антитело или антигенсвязывающий фрагмент по настоящему изобретению содержит VH, содержащий или состоящий из аминокислотной последовательности согласно SEQ ID NO:113, и VL, содержащий или состоящий из аминокислотной последовательности согласно SEQ ID NO:168, и связывается с S-белком RBD SARS-CoV-2 с EC50 от около 9 до около 23 нг/мл. В некоторых вариантах осуществления антитело или антигенсвязывающий фрагмент по настоящему изобретению содержит VH, содержащий или состоящий из аминокислотной последовательности согласно SEQ ID NO:129, и VL, содержащий или состоящий из аминокислотной последовательности согласно SEQ ID NO:168, и связывается с S-белком RBD SARS-CoV-2 с EC50 от около 8 до около 22 нг/мл. В некоторых вариантах осуществления антитело или антигенсвязывающий фрагмент по настоящему изобретению содержит VH, содержащий или состоящий из аминокислотной последовательности согласно SEQ ID NO:119, и VL, содержащий или состоящий из аминокислотной последовательности согласно SEQ ID NO:168, и связывается с S-белком RBD SARS-CoV-2 с EC50 от около 8 до около 22 нг/мл. В некоторых вариантах осуществления антитело или антигенсвязывающий фрагмент по настоящему изобретению содержит VH, содержащий или состоящий из аминокислотной последовательности согласно SEQ ID NO:172, и VL, содержащий или состоящий из аминокислотной последовательности согласно SEQ ID NO:168, и связывается с S-белком RBD SARS-CoV-2 с EC50 от около 7 до около 19 нг/мл.
В некоторых вариантах осуществления антитело или антигенсвязывающий фрагмент по настоящему изобретению является моноспецифическим (например, связывается с одним эпитопом) или является полиспецифическим (например, связывается с несколькими эпитопами и/или молекулами-мишенями). Антитела и антигенсвязывающие фрагменты могут быть сконструированы в различных форматах. Примерные форматы антител, описанные в Spiess и др., Mol. Immunol. 67(2):95 (2015), и в Brinkmann и Kontermann, mAb 9(2): 182-212 (2017), форматы и способы их получения включены в настоящий документ посредством ссылки и включают, например, активаторы биспецифических Т-клеток (BiTE), DART, сборки "выступ-во-впадину" (KIH), сборки scFv-CH3-KIH, KIH антитела с общей легкой цепью, TandAbs, тройные тела, мини-тела TriBi, Fab-scFv, scFv-CH-CL-scFv, F(ab') 2-scFv2, четырехвалентные HCabs, интратела, CrossMabs, Fabs двойного действия (DAFs) (два в одном или четыре в одном), DutaMabs, DT-IGG, пары зарядов, Fab-arm Exchange, SEEDbodies, Triomabs, LZ-Y сборки, Fcabs, κλ-тела или ортотела Fabs, DVDgs (например, патент США № 8,258,268, форматы, которые включены в настоящий документ полностью посредством ссылки), IgFv- IgG(H)-scFv, scFv-(H)IgG, IgG(L)-scFv, scFv-(L)IgG, IgG(L,H)-Fv, IgG(H)-V, V(H)-IgG, IgG(L)-V, V(L)-IgG, KIH IgG-scFab, 2scFv-IgG, IgG-2scFv, scFv4-Ig, Zybody, и DVI-IgG, так же так называемые, как и FIT-Ig (например, публикация РСТ № WO 2015/103072, форматы которых полностью включены в настоящий документ посредством ссылки), так называемые форматы WuxiBody (например, публикация РСТ № WO 2019/057122, форматы которых полностью включены в настоящий документ посредством ссылки), и так называемые форматы Ig In-Elbow-Insert (вставка в изгиб) (IEI-Ig; например, публикации PCT № WO 2019/024979 и WO 2019/025391, форматы которых полностью включены в настоящий документ посредством ссылки).
В некоторых вариантах осуществления указанное антитело или антигенсвязывающий фрагмент содержит два или более доменов VH, два или более доменов VL или и то, и другое (т.е., два или более доменов VH и два или более доменов VL). В частных вариантах осуществления антигенсвязывающий фрагмент содержит формат (от N-конца к C-концу) VH-линкер-VL-линкер-VH-линкер-VL, где две последовательности VH могут быть одинаковыми или разными, и две последовательности VL могут быть одинаковыми или разными. Такие связанные scFv могут включать любую комбинацию доменов VH и VL, предназначенных для связывания с данной мишенью, и в форматах, содержащих два или более VH и/или два или более VL, могут быть связаны один, два или более различных эпитиопов или антигенов. Следует понимать, что форматы, включающие несколько антигенсвязывающих доменов, могут включать последовательности VH и/или VL в любой комбинации или ориентации. Например, антигенсвязывающий фрагмент может содержать формат VL-линкер-VH-линкер-VL-линкер-VH, VH-линкер-VL-линкер-VL-линкер-VH или VL-линкер-VH-линкер-VH-линкер-VL.
Моноспецифические или полиспецифические антитела или антигенсвязывающие фрагменты по настоящему изобретению могут содержать любую комбинацию последовательностей VH и VL и/или любую комбинацию последовательностей CDRH1, CDRH2, CDRH3, CDRL1, CDRL2 и CDRL3, описанных в настоящем документе. В некоторых вариантах осуществления антитело или антигенсвязывающий фрагмент содержит последовательность VH, представленную в SEQ ID NO:105 или SEQ ID NO:113, и последовательность VL, представленную в SEQ ID NO: 168. В некоторых вариантах осуществления антитело или антигенсвязывающий фрагмент содержит последовательность CDRH1, представленную в SEQ ID NO: 106, последовательность CDRH2, представленную в SEQ ID NO: 107 или 121, последовательность CDRH3, представленную в SEQ ID NO: 108, последовательность CDRL1, представленную в SEQ ID NO: 169, последовательность CDRL2, представленную в SEQ ID NO: 170, и последовательность CDRL3, представленную в SEQ ID NO: 171. В некоторых вариантах осуществления антитело или антигенсвязывающий фрагмент содержит аминокислотную последовательность, представленную в любой из SEQ ID NO: 222-225 или 230-233. Биспецифическое или полиспецифическое антитело или антигенсвязывающий фрагмент может, в некоторых вариантах осуществления изобретения, содержать один, два или более антигенсвязывающих доменов (например, VH и VL) согласно настоящему описанию. Могут присутствовать два или более связывающих домена, которые связываются с одним и тем же или другим эпитопом SARS-CoV-2, и биспецифическое или полиспецифическое антитело или антигенсвязывающий фрагмент, предложенные в настоящем документе, могут, в некоторых вариантах осуществления, содержать дополнительный связывающий домен SARS-CoV-2 и/или могут содержать связывающий домен, который вообще связывается с другим антигеном или патогеном.
В любом из раскрытых в настоящем документе вариантов осуществления антитело или антигенсвязывающий фрагмент могут быть полиспецифическими; например, биспецифическими, триспецифическими или т. п.
В некоторых вариантах осуществления антитело или его антигенсвязывающий фрагмент содержит: (i) первый VH и первый VL; и (ii) второй VH и второй VL, где первый VH и второй VH являются различными и каждый независимо содержит аминокислотную последовательность, имеющую по меньшей мере 85% (т.е.85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100%) идентичности с аминокислотной последовательностью, представленной в любой из SEQ ID NO: 1, 9-15, 23, 24, 27-46, 55, 63, 79, 87, 95, 103, 105, 113-120, 129-146, 155, 172, 176-178, 194, 196, 198, 200, 202 и 239, и где первый VL и второй VL являются различными, и каждая независимо содержит аминокислотную последовательность, имеющую по меньшей мере 85% (т.е.85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100%) идентичности с аминокислотной последовательностью, представленной в любой из SEQ ID NO: 5, 47-50, 59, 67, 71, 72, 75, 76, 83, 91, 99, 109, 147-150, 159, 168, 182, 190, 234 и 243, и где первая VH и первая VL вместе образуют первый антигенсвязывающий сайт, и где второй VH и второй VL вместе образуют второй антигенсвязывающий сайт.
В некоторых вариантах осуществления антитело или его антигенсвязывающий фрагмент содержит полипептид Fc или его фрагмент. "Fc" содержит карбоксиконцевые части (т.е. домены IgG CH2 и CH3 ) обеих цепей антитела H, удерживаемых вместе дисульфидами. "Эффекторные функции" антитела относятся к биологической активности, относящейся к Fc-области (Fc-области нативной последовательности или Fc-области варианта аминокислотной последовательности) антитела, и варьируются в зависимости от изотипа антитела. Примеры эффекторных функций антител включают: связывание C1q и комплемент-зависимую цитотоксичность; связывание с рецептором Fc; антитело-зависимую клеточно-опосредованную цитотоксичность (ADCC); фагоцитоз; подавление рецепторов поверхности клетки (например, рецептора B-клеток); и активацию B-клеток. Как обсуждается в настоящем документе, модификации (например, аминокислотные замены) могут быть сделаны в Fc-домене с целью модификации (например, улучшения, уменьшения или удаления) одной или более функциональных групп Fc-содержащего полипептида (например, антитела по настоящему изобретению). Такие функции включают, например, связывание с Fc-рецептором (FcR), модуляцию периода полужизни антитела (например, путем связывания с FcRn), функцию ADCC, связывание с белком A, связывание с белком G и связывание с комплементом. Аминокислотные модификации, которые модифицируют (например, улучшают, уменьшают или удаляют) Fc-функции, включают, например, TT250Q/M428L, M252Y/S254T/T256E, H433K/N434F, M428L/N434S, E233P/L234V/L235A/G236 + A327G/A330S/P331S, E333A, S239D/A330L/I332E, P257I/Q311, K326W/E333S, S239D/I332E/G236A, N297Q, K322A, S228P, L235E + E318A/K320A/K322A, L234A/L235A (также называемые в настоящем документе «LALA») и мутации L234A/L235A/P329G, которые обобщены и аннотированы в «Сконструированных Fc-областей», опубликованных InvenGo (2011) и доступны в Интернете по адресу invivogenPreview.com/DFre/review-Eng-Egene-Fegogen-in-in.pdf.utm_source=review&utm_medium=pdf&utm_campaign=review&utm_content=Engineered-Fc-Regions, и включены в настоящий документ посредством ссылки. Если из контекста не следует иное, аминокислотные остатки Fc пронумерованы в настоящем документе в соответствии с системой нумерации EU.
Например, для активации каскада комплемента белковый комплекс C1q может связываться по меньшей мере с двумя молекулами IgG1 или одной молекулой IgM, когда к антигенной мишени присоединена (Ward, E. S., и Ghetie, V., Ther. Immunol. 2 (1995) 77-94). Burton, D. R., раскрыл (Mol. Immunol. 22 (1985) 161-206), что область тяжелой цепи, содержащая аминокислотные остатки в положении 318 до 337, участвует в фиксации комплемента. Duncan, A. R., и Winter, G. (Nature 332 (1988) 738-740), используя сайт-специфический мутагенез, сообщили, что Glu318, Lys320 и Lys322 образуют сайт связывания с C1q. Роль остатков Glu318, Lys320 и Lys 322 в связывании C1q была подтверждена способностью короткого синтетического пептида, содержащего эти остатки, ингибировать опосредованный комплементом лизис.
Например, связывание FcR может быть опосредовано взаимодействием Fc-фрагмента (антитела) с Fc-рецепторами (FcR), которые представляют собой специализированные рецепторы клеточной поверхности на клетках, включая гемопоэтические клетки. Fc-рецепторы принадлежат к суперсемейству иммуноглобулинов и, как показано, опосредуют как удаление патогенов, покрытых антителами, путем фагоцитоза иммунных комплексов, так и лизис эритроцитов и различных других клеточных мишеней (например, опухолевых клеток), покрытых соответствующим антителом, посредством антителозависимой клеточной цитотоксичности (ADCC; Van de Winkel, J. G., и Anderson, C. L., J. Leukoc. Biol. 49 (1991) 511-524). FcR определяются их специфичностью для классов иммуноглобулинов; Fc-рецепторы для антител IgG называются FcγR, для IgE - FcεR, для IgA - FcαR и так далее, а неонатальные Fc-рецепторы называются FcRn. Связывание с Fc-рецептором описано, например, в Ravetch, J. V., и Kinet, J. P., Annu. Rev. Immunol. 9 (1991) 457-492; Capel, P. J., et al., Immunomethods 4 (1994) 25-34; de Haas, M., и др., J Lab. Clin. Med. 126 (1995) 330-341; и Gessner, J. E., и др., Ann. Hematol. 76 (1998) 231-248.
Перекрестное сшивание рецепторов Fc-доменом нативных антител IgG (FcγR) запускает широкий спектр эффекторных функций, включая фагоцитоз, антителозависимую клеточную цитотоксичность и высвобождение воспалительных медиаторов, а также клиренс иммунного комплекса и регуляцию продукции антител. В настоящем документе рассматриваются Fc-фрагменты, обеспечивающие перекрестное сшивание рецепторов (например, FcγR). У человека к настоящему времени были охарактеризованы три класса FcγR, которые представляют собой: (i) FcγRI (CD64), который связывает мономерный IgG с высокой аффинностью и экспрессируется на макрофагах, моноцитах, нейтрофилах и эозинофилах; (ii) FcγRII (CD32), который связывает комплексный IgG со средним или низким сродством, широко экспрессируется, в частности, на лейкоцитах, который, как полагают, является центральным игроком в опосредованном антителом иммунитете, и который может быть разделен на FcγRIIA, FcγRIIB и FcγRIIC, которые выполняют различные функции в иммунной системе, но связываются с подобной низкой аффиностью с IgG-Fc, и эктодомены этих рецепторов являются высоко гомологичными; и (iii) FcγRIII (CD16), который связывает IgG со средним или низким сродством и был обнаружен в двух формах: FcγRIIIA, который был обнаружен на NK-клетках, макрофагах, эозинофилах и некоторых моноцитах и T-клетках, и, как полагают, опосредует ADCC и FcγRIIIB, который высоко экспрессируется на нейтрофилах.
FcγRIIA обнаружен во многих клетках, участвующих в уничтожении (например, макрофагах, моноцитах, нейтрофилах), и, по-видимому, способен активировать процесс уничтожения. FcγRIIB, по-видимому, играет роль в ингибирующих процессах и обнаруживается на В-клетках, макрофагах и на тучных клетках и эозинофилах. Важно отметить, что 75% всех FcγRIIB обнаружено в печени (Ganesan, L. P. и др., 2012: "FcγRIIb on liver sinusoidal endothelium clears small immune complexes," Journal of Immunology 189: 4981-4988). FcγRIIB обильно экспрессируется на синусоидальном эндотелии печени, называемом LSEC, а в клетках Купфера в печени и LSEC являются основным местом клиренса малых иммунных комплексов (Ganesan, L. P. и др., 2012: FcγRIIb на синусоидальном эндотелии печени очищает малые иммунные комплексы. Journal of Immunology 189: 4981-4988).
В некоторых вариантах осуществления антитела, описанные в данном документе, и их антигенсвязывающие фрагменты содержат полипептид Fc или его фрагмент для связывания с FcγRIIb, в частности, область Fc, такую как, например, антитела IgG-типа. Кроме того, можно сконструировать Fc-фрагмент для усиления связывания FcγRIIB путем введения мутаций S267E и L328F, как описано Chu, S.Y. и др., 2008: Inhibition of B cell receptor-mediated activation of primary human B cells by coengagement of CD19 and FcgammaRIIb with Fc-engineered antibodies. Molecular Immunology 45, 3926-3933. Таким образом, клиренс иммунных комплексов может быть увеличен (Chu, S., и др., 2014: Accelerated Clearance of IgE In Chimpanzees Is Mediated By Xmab7195, An Fc-Engineered Antibody With Enhanced Affinity For Inhibitory Receptor FcγRIIb. Am J Respir Crit, American Thoracic Society International Conference Abstracts). В некоторых вариантах осуществления антитела по настоящему изобретению или их антигенсвязывающие фрагменты содержат сконструированный фрагмент Fc с мутациями S267E и L328F, в частности, как описано в Chu, S.Y. и др., 2008: Inhibition of B cell receptor-mediated activation of primary human B cells by coengagement of CD19 and FcgammaRIIb with Fc-edineered antibodies. Molecular Immunology 45, 3926-3933.
На В-клетках FcγRIIB может функционировать для подавления дальнейшей продукции иммуноглобулина и переключения изотипа на, например, класс IgE. Считается, что на макрофагах FcγRIIB ингибирует фагоцитоз, опосредованный через FcγRIIA. На эозинофилах и тучных клетках В-форма может способствовать подавлению активации этих клеток посредством связывания IgE с отдельным рецептором.
Что касается связывания FcγRI, модификация в нативном IgG по меньшей мере одного из E233-G236, P238, D265, N297, A327 и P329 снижает связывание с FcγRI. Остатки IgG2 в положениях 233-236, замещенные в соответствующие положения IgG1 и IgG4, снижают связывание IgG1 и IgG4 с FcγRI в 103 раза и устраняют моноцитарный ответ человека на сенсибилизированные антителами эритроциты (Armour, K. L., и др. Eur. J. Immunol. 29 (1999) 2613-2624).
Что касается связывания FcγRII, то обнаружено снижение связывания с FcγRIIA, например, для мутации IgG по меньшей мере одного из E233-G236, P238, D265, N297, A327, P329, D270, Q295, A327, R292 и K414.
Двумя аллельными формами FcγRIIA человека являются вариант "H131", который связывается с Fc IgG1 с высокой аффинностью, и вариант "R131", который связывается с Fc IgG1 с низкой аффинностью. См., например, Bruhns и др., Blood 113:3716-3725 (2009).
Что касается связывания FcγRIII, то обнаружено снижение связывания с FcγRIIIA, например, для мутации по меньшей мере одного из E233-G236, P238, D265, N297, A327, P329, D270, Q295, A327, S239, E269, E293, Y296, V303, A327, K338 и D376. Картирование сайтов связывания на IgG1 человека для Fc-рецепторов, вышеупомянутые сайты мутации и способы измерения связывания с FcγRI и FcγRIIA описаны в Shields, R. L., и др., J. Biol. Chem. 276 (2001) 6591-6604.
Двумя аллельными формами FcγRIIIA человека являются вариант "F158", который связывается с Fc IgG1 с низкой аффинностью, и вариант "V158", который связывается с Fc IgG1 с высокой аффинностью. См., например, Bruhns и др., Blood 113:3716-3725 (2009).
Что касается связывания с FcγRII, то две области нативного Fc IgG, по-видимому, участвуют во взаимодействии между FcγRII и IgG, а именно (i) нижний шарнирный сайт Fc IgG, в частности, аминокислотные остатки L, L, G, G (234 - 237, нумерация EU), и (ii) смежная область домена CH2 Fc IgG, в частности, петля и цепи в верхнем домене CH2, прилегающие к нижней шарнирной области, например, в области P331 (Wines, B.D., и др., J. Immunol. 2000; 164: 5313 - 5318). Кроме того, FcγRI, по-видимому, связывается с одним и тем же сайтом на Fc IgG, тогда как FcRn и белок A связываются с другим сайтом на Fc IgG, который, по-видимому, находится на границе раздела CH2-CH3 (Wines, B.D., и др., J. Immunol. 2000; 164: 5313 - 5318).
Также рассматриваются мутации, которые увеличивают аффинность связывания полипептида Fc или его фрагмента по настоящему изобретению с (т.е., одним или более) рецептором Fcγ (например, по сравнению с эталонным полипептидом Fc или его фрагментом или содержащим его, который не содержит мутацию (мутации); например, полипептидом Fc дикого типа или его фрагментом (например, того же изотипа, что и полипептид Fc или его фрагмент, который содержит мутацию или мутации) или полипептидом Fc или его фрагментом, который иным образом идентичен или по существу идентичен полипептиду Fc или его фрагменту, который содержит мутацию или мутации). См., например, Delillo и Ravetch, Cell 161(5):1035-1045 (2015) и Ahmed и др., J. Struc. Biol. 194(1):78 (2016), мутации Fc и методики которых включены в настоящий документ посредством ссылки.
В любом из раскрытых в настоящем документе вариантов осуществления антитело или антигенсвязывающий фрагмент могут содержать полипептид Fc или его фрагмент, содержащий мутацию, выбранную из G236A; S239D; A330L; и I332E; или комбинацию, включающую любые две или более из них; например, S239D/I332E; S239D/A330L/I332E; G236A/S239D/I332E; G236A/A330L/I332E (также называемую в настоящем документе "GAALIE"); или G236A/S239D/A330L/I332E. В некоторых вариантах осуществления изобретения полипептид Fc или его фрагмент не содержит S239D. В некоторых вариантах осуществления полипептид Fc или его фрагмент содержит S в положении 239.
В некоторых вариантах осуществления полипептид Fc или его фрагмент может содержать или состоять по меньшей мере из части полипептида Fc или его фрагмента, которая участвует в связывании со связыванием FcRn. В некоторых вариантах осуществления полипептид Fc или его фрагмент содержит одну или более аминокислотных модификаций, которые улучшают аффинность связывания с (например, усиливают связывание с) FcRn (например, при рН около 6,0) и, таким образом, в некоторых вариантах осуществления продлеваютпериод полувыведения молекулы in vivo, содержащей полипептид Fc или его фрагмент (например, по сравнению с эталонным (например, дикого типа) полипептидом Fc или его фрагментом или антителом, которое в противном случае является таким же, но не содержит модификацию (модификации)). В некоторых вариантах осуществления полипептид Fc или его фрагмент содержит или получен от Fc IgG, и мутация, продлевающая период полувыведения, включает одну любую одну или более из: M428L; N434S; N434H; N434A; N434S; M252Y; S254T; T256E; T250Q; P257I Q311I; D376V; T307A; E380A (нумерация EU). В некоторых вариантах осуществления мутация, продлевающая период полувыведения, включает M428L/N434S (также называемая в данном документе "MLNS" или "LS"). В некоторых вариантах осуществления мутация, продлевающая период полувыведения, включает M252Y/S254T/T256E. В некоторых вариантах осуществления мутация, продлевающая период полувыведения, включает T250Q/M428L. В некоторых вариантах осуществления мутация, продлевающая период полувыведения, включает P257I/Q311I. В некоторых вариантах осуществления мутация, продлевающая период полувыведения, включает P257I/N434H. В некоторых вариантах осуществления мутация, продлевающая период полувыведения, включает D376V/N434H. В некоторых вариантах осуществления мутация, продлевающая период полувыведения, включает T307A/E380A/N434A.
В некоторых вариантах осуществления антитело или антигенсвязывающий фрагмент включает Fc-фрагмент, который содержит замены M428L/N434S. В некоторых вариантах осуществления антитело или антигенсвязывающий фрагмент включает полипептид Fc или его фрагмент, который содержит замены мутаций G236A/A330L/I332E. В некоторых вариантах осуществления антитело или антигенсвязывающий фрагмент содержит (например, IgG) Fc-фрагмент, который содержит мутацию G236A, мутацию A330L и мутацию I332E (GAALIE) и не содержит мутацию S239D (например, содержит нативный S в положении 239). В частных вариантах осуществления антитело или антигенсвязывающий фрагмент включает полипептид Fc или его фрагмент, который содержит мутации по типу замены: M428L/N434S и G236A/A330L/I332E, и необязательно не содержит S239D. В некоторых вариантах осуществления антитело или его антигенсвязывающий фрагмент включает полипептид Fc или его фрагмент, который содержит мутации по типу замены: M428L/N434S и G236A/S239D/A330L/I332E.
В некоторых вариантах осуществления антитело или антигенсвязывающий фрагмент содержит мутацию, которая изменяет гликозилирование, где мутация, которая изменяет гликозилирование, включает N297A, N297Q или N297G, и/или антитело или антигенсвязывающий фрагмент частично или полностью агликозилировано и/или частично или полностью афукозилировано. Известны линии клеток-хозяев и способы получения частично или полностью агликозилированных или частично или полностью афукозилированных антител и антигенсвязывающих фрагментов (см., например, публикацию РСТ № WO 2016/181357; Suzuki и др. Clin. Cancer Res. 13(6):1875-82 (2007); Huang и др. MAbs 6:1-12 (2018)).
В некоторых вариантах осуществления антитело или антигенсвязывающий фрагмент способны вызывать непрерывную защиту in vivo у субъекта, даже если у субъекта не могут быть обнаружены детектируемые уровни антитела или антигенсвязывающего фрагмента (то есть, если антитело или антигенсвязывающий фрагмент было удалено от субъекта после введения). Такая защита упоминается в настоящем документе как вакцинальный эффект. Не ограничиваясь теорией, полагают, что дендритные клетки могут интернализировать комплексы антитела и антигена и после этого индуцировать или способствовать эндогенному иммунному ответу против антигена. В некоторых вариантах осуществления антитело или антигенсвязывающий фрагмент содержит одну или более модификаций, таких как, например, мутации в Fc, содержащие G236A, A330L и I332E, которые способны активировать дендритные клетки, которые могут индуцировать, например, иммунитет Т-клеток к антигену.
В любом из раскрытых в настоящем документе вариантов осуществления антитело или его антигенсвязывающий фрагмент содержит полипептид Fc или его фрагмент, включая CH2 (или его фрагмент, CH3 (или его фрагмент) или CH2 и CH3, где CH2, CH3 или оба могут быть любого изотипа и могут содержать аминокислотные замены или другие модификации по сравнению с соответствующим CH2 или CH3 дикого типа, соответственно. В некоторых вариантах осуществления полипептид Fc по настоящему изобретению содержит два полипептида CH2-CH3, которые ассоциируются с образованием димера.
В любом из раскрытых в настоящем документе вариантов осуществления антитело или антигенсвязывающий фрагмент может быть моноклональным. Термин "моноклональное антитело" (mAb) в настоящем документе относится к антителу, полученному из популяции по существу однородных антител, то есть отдельные антитела в составе популяции являются идентичными, за исключением возможных естественно присутствующих мутаций, которые могут присутствовать в небольших количествах. Моноклональные антитела являются высокоспецифичными, поскольку они направлены против одной области детерминанты. Кроме того, по сравнению с препаратами поликлональных антител, содержащими различные антитела, направленные против различных эпитопов, каждое моноклональное антитело направлено против одного эпитопа антигена. Кроме специфичности, моноклональные антитела обладают преимуществом, заключающимся в том, что их можно синтезировать в виде, не загрязненном другими антителами. Определение «моноклональный» не следует толковать как требующий продуцирования антитела каким-либо конкретным способом. Например, моноклональные антитела, пригодные для применения в настоящем изобретении, могут быть получены с помощью гибридомной методики, впервые описанной Kohler и др., Nature 256:495 (1975), или могут быть получены с использованием способов рекомбинантной ДНК в клетках бактерий, эукариотических животных или растений (см., например, патент США № 4816567). Моноклональные антитела также могут быть выделены из библиотек фаговых антител с использованием методик, описанных в Clackson и др., Nature, 352:624-628 (1991) и Marks и др., J. Mol. Biol., 222:581-597 (1991), например. Моноклональные антитела также могут быть получены с использованием способов, описанных в публикации РСТ № WO 2004/076677A2.
Антитела и антигенсвязывающие фрагменты согласно настоящему изобретению включают «химерные антитела», в которых часть тяжелой и/или легкой цепи идентична или гомологична соответствующим последовательностям антител, полученных от конкретного вида или принадлежащих к конкретному классу или подклассу антител, в то время как остальная часть цепи (цепей) идентична или гомологична соответствующим последовательностям антител, полученных от другого вида или принадлежащих к другому классу или подклассу антител, а также фрагментам таких антител, при условии, что они проявляют желаемую биологическую активность (см. патент США № 4,816,567; 5,530,101 и 7,498,415; и Morrison и др., Proc. Natl. Acad. Sci. USA, 81:6851-6855 (1984)). Например, химерные антитела могут содержать человеческие и нечеловеческие остатки. Кроме того, химерные антитела могут содержать остатки, которые не обнаружены в реципиентном антителе или в донорском антителе. Эти модификации сделаны для дополнительного улучшения характеристик антител. Для получения дополнительной информации см. Jones и др., Nature 321:522-525 (1986); Riechmann и др., Nature 332:323-329 (1988); и Presta, Curr. Op. Struct. Biol.2:593-596 (1992). Химерные антитела также включают приматизированные и гуманизированные антитела.
«Гуманизированное антитело» обычно считают антителом человека, которое содержит один или более аминокислотных остатков, введенных в него из источника, который не является человеком. Эти аминокислотные остатки нечеловеческого происхождения обычно берут из вариабельного домена. Гуманизация может быть выполнена по методу Winter и сотрудников (Jones и др., Nature, 321:522-525 (1986); Reichmann и др., Nature, 332:323-327 (1988); Verhoeyen и др.., Science, 239:1534-1536 (1988)), путем замены нечеловеческих вариабельных последовательностей на соответствующие последовательности человеческого антитела. Соответственно, такие «гуманизированные» антитела представляют собой химерные антитела (патент США № 4,816,567; 5,530,101 и 7,498,415), где по существу менее, чем интактный вариабельный домен человека, замещен соответствующей последовательностью из вида, не являющегося человеком. В некоторых случаях «гуманизированное» антитело представляет собой антитело, которое продуцируется нечеловеческой клеткой или клеткой животного, и содержит человеческие последовательности, например, домены HC.
«Антитело человека» представляет собой антитело, содержащее только последовательности, которые присутствуют в антителе, продуцируемом человеком. Однако в данном контексте человеческие антитела могут содержать остатки или модификации, не обнаруженные в природном человеческом антителе (например, антителе, выделенном из человека), включая те модификации и варианты последовательностей, которые описаны в данном документе. Они, как правило, выполняются для дополнительного улучшения или повышения характеристик антител. В некоторых случаях антитела человека продуцируются трансгенными животными. Например, см. патент США № 5,770,429; 6,596,541 и 7,049,426.
В некоторых вариантах осуществления антитело или антигенсвязывающий фрагмент согласно настоящему изобретению является химерным, гуманизированным или человеческим.
Полинуклеотиды, векторы и клетки-хозяева
В другом аспекте настоящее изобретение относится к выделенным полинуклеотидам, которые кодируют любое из описанных в настоящем документе антител или их антигенсвязывающий фрагмент, или их часть (например, CDR, VH, VL, тяжелую цепь или легкую цепь). В некоторых вариантах осуществления полинуклеотид является кодон-оптимизированным для экспрессии в клетке-хозяине. После того, как кодирующая последовательность известна или идентифицирована, оптимизация кодонов может быть выполнена с использованием известных методик и инструментов, например, с использованием инструмента GenScript® OptimiumGene™ или Gen Synthesis by GeneArt® (ThermoFisher); см. также Scholten и др.., Clin. Immunol. 119:135, 2006). Кодон-оптимизированные последовательности включают последовательности, которые частично кодон-оптимизированы (то есть, один или множество кодонов оптимизированы для экспрессии в клетке-хозяине), и те, которые полностью кодон-оптимизированы.
Следует также понимать, что полинуклеотиды, кодирующие антитела и антигенсвязывающие фрагменты по настоящему изобретению, могут обладать различными нуклеотидными последовательностями, при этом все еще кодируя одно и то же антитело или антигенсвязывающий фрагмент из-за, например, вырожденности генетического кода, сплайсинга и т. п.
В некоторых вариантах осуществления полинуклеотид содержит полинуклеотид, обладающий по меньшей мере 50% (т.е. 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100%) идентичностью полинуклеотидной последовательности согласно любой одной или более из SEQ ID NO:186-189, 191-192, 238, 247, 248-250, 254-255 и 257-262.
Следует понимать, что в некоторых вариантах осуществления полинуклеотид, кодирующий антитело или антигенсвязывающий фрагмент, содержится в полинуклеотиде, который содержит другие последовательности и/или признаки, например, экспрессии антитела или антигенсвязывающего фрагмента в клетке-хозяине. Примерные признаки включают последовательность промотора, последовательность полиаденилирования, последовательность, которая кодирует сигнальный пептид (например, расположенный на N-конце экспрессированной тяжелой цепи антитела или легкой цепи), или тому подобное. Соответственно, в некоторых вариантах осуществления полинуклеотид дополнительно содержит полинуклеотидную последовательность, имеющую по меньшей мере 50% идентичности с, содержащей или состоящей из полинуклеотидной последовательности, представленной в любой из SEQ ID NO:251-253 и 263. В некоторых вариантах осуществления полинуклеотид содержит последовательность, которая кодирует сигнальный пептид (также называемый лидерной последовательностью), имеющий по меньшей мере 90% идентичности с, содержащей или состоящей из аминокислотной последовательности, представленной в SEQ ID NO: 256 или SEQ ID NO: 264.
В любом из раскрытых в настоящем документе вариантов осуществления полинуклеотид может содержать дезоксирибонуклеиновую кислоту (ДНК) или рибонуклеиновую кислоту (РНК). В некоторых вариантах осуществления изобретения РНК включает матричную РНК (мРНК).
Также предложены векторы, где векторы содержат или включают полинуклеотид, описанный в настоящем документе (например, полинуклеотид, кодирующий антитело или антигенсвязывающий фрагмент, который связывается с SARS-CoV-2). Вектор может содержать любой один или более из векторов, описанных в настоящем документе. В частных вариантах осуществления предложен вектор, который содержит конструкцию ДНК-плазмиды, кодирующую антитело или антигенсвязывающий фрагмент, или их часть (например, так называемое "DMAb"; см., например, Muthumani и др., J Infect Dis. 214(3):369-378 (2016); Muthumani и др.., Hum Vaccin Immunother 9:2253-2262 (2013)); Flingai и др., Sci Rep. 5:12616 (2015); и Elliott и др., NPJ Vaccines 18 (2017), где конструкции ДНК, кодирующие антитело, и родственные способы применения, включая введения того же самого, включены в настоящий документ в качестве ссылки).). В некоторых вариантах осуществления конструкция ДНК-плазмиды содержит одну открытую рамку считывания, кодирующую тяжелую цепь и легкую цепь (или VH и VL) антитела или антигенсвязывающего фрагмента, где последовательность, кодирующая тяжелую цепь, и последовательность, кодирующая легкую цепь, необязательно разделены полинуклеотидом, кодирующим сайт расщепления протеазой, и/или полинуклеотидом, кодирующим саморасщепляющийся пептид. В некоторых вариантах осуществления заместительные компоненты антитела или антигенсвязывающего фрагмента кодируются полинуклеотидом, содержащимся в одной плазмиде. В других вариантах осуществления заместительные компоненты антитела или антигенсвязывающего фрагмента кодируются полинуклеотидом, содержащимся в двух или более плазмидах (например, первая плазмида содержит полинуклеотид, кодирующий тяжелую цепь, VH или VH+CH, а вторая плазмида содержит полинуклеотид, кодирующий родственную легкую цепь, VL или VL+CL). В некоторых вариантах осуществления одна плазмида содержит полинуклеотид, кодирующий тяжелую цепь и/или легкую цепь из двух или более антител или антигенсвязывающих фрагментов согласно настоящему изобретению. Иллюстративный вектор экспрессии представляет собой pVax1, доступный от Invitrogen®. ДНК-плазмида по настоящему изобретению может быть доставлена субъекту, например, с помощью электропорации (например, внутримышечной электропорации) или с соответствующим составом (например, гиалуронидазой). В некоторых вариантах осуществления вектор согласно настоящему изобретению содержит нуклеотидную последовательность, кодирующую сигнальный пептид. Сигнальный пептид может присутствовать или не присутствовать (например, может быть ферментативно отщеплен от) на зрелом антителе или антигенсвязывающем фрагменте. В некоторых вариантах осуществления сигнальный пептид кодируется нуклеотидной последовательностью, представленной в SEQ ID NO: 252 или SEQ ID NO: 263, и/или сигнальный пептид содержит или состоит из аминокислотной последовательности, представленной в SEQ ID NO:256 или SEQ ID NO: 264. В некоторых вариантах осуществления вектор согласно настоящему изобретению содержит сигнальную последовательность полиаденилирования. В некоторых вариантах осуществления сигнальная последовательность полиаденилирования содержит или состоит из нуклеотидной последовательности, представленной в SEQ ID NO: 253.
В некоторых вариантах осуществления вектор согласно настоящему изобретению содержит промотор CMV. В некоторых вариантах осуществления промотор содержит или состоит из нуклеотидной последовательности, представленной в SEQ ID NO: 251.
В дополнительном аспекте настоящее изобретение также относится к клетке-хозяину, экспрессирующей антитело или антигенсвязывающий фрагмент согласно настоящему изобретению; или содержащей или включающей вектор или полинуклеотид согласно настоящему изобретению.
Примеры таких клеток включают, но не ограничиваются ими, эукариотические клетки, например, дрожжевые клетки, клетки животных, клетки насекомых, клетки растений; и прокариотические клетки, включая E. coli. В некоторых вариантах осуществления изобретения клетки представляют собой клетки млекопитающих. В некоторых таких вариантах осуществления клетки представляют собой клеточную линию млекопитающих, такую как клетки CHO (например, клетки DHFR- CHO (Urlaub и др., PNAS 77:4216 (1980)), эмбриональные клетки почек человека (например, клетки HEK293T), клетки PER.C6, клетки Y0, клетки Sp2/0. Клетки NS0, клетки печени человека, например, клетки Hepa RG, клетки миеломы или клетки гибридомы. Другие примеры линий клеток-хозяев млекопитающих включают клетки Сертоли мыши (например, клетки TM4); линию CV1 почки обезьяны, трансформированную SV40 (COS-7); клетки почки новорожденного хомяка (BHK); клетки почки африканской зеленой мартышки (VERO-76); клетки почки обезьяны (CV1); клетки карциномы шейки матки человека (HELA); клетки легкого человека (W138); клетки печени человека (Hep G2); клетки почки собаки (MDCK); клетки печени крысы Buffalo (BRL 3A); опухоль молочной железы мыши (MMT 060562); клетки TRI; клетки MRC 5; и клетки FS4. Линии клеток-хозяев млекопитающих, подходящие для продукции антител, также включают те, которые описаны, например, в Yazaki и Wu, Methods in Molecular Biology, Vol. 248 (B. K. C. Lo, ed., Humana Press, Totowa, N.J.), pp. 255-268 (2003).
В некоторых вариантах осуществления клетка-хозяин представляет собой прокариотическую клетку, такую как E. coli. Экспрессия пептидов в прокариотических клетках, таких как E. coli , хорошо известна (см., например, Pluckthun, A. Bio/Technology 9:545-551 (1991). Например, антитела можно продуцировать в бактериях, в частности, если гликозилирование и Fc-эффекторные функции не нужны. Экспрессию фрагментов антител и полипептидов в бактериях см., например, в патенте США № 5,648,237; 5,789,199; и 5,840,523.
В частных вариантах осуществления клетка может быть трансфицирована вектором в соответствии с настоящим описанием с вектором экспрессии. Термин "трансфекция" относится к введению молекул нуклеиновых кислот, таких как молекулы ДНК или РНК (например, мРНК), в клетки, такие как эукариотические клетки. В контексте настоящего описания термин «трансфекция» охватывает любой известный специалисту способ введения молекул нуклеиновых кислот в клетки, например, в эукариотические клетки, в том числе в клетки млекопитающих. Такие способы включают, например, электропорацию, липофекцию, например, на основе катионных липидов и/или липосом, осаждение фосфатом кальция, трансфекцию на основе наночастиц, трансфекцию на основе вируса или трансфекцию на основе катионных полимеров, таких как DEAE-декстран или полиэтиленимин и т.д. В некоторых вариантах осуществления введение является невирусным.
Кроме того, клетки-хозяева согласно настоящему изобретению могут быть стабильно или временно трансфицированы вектором согласно настоящему изобретению, например, для экспрессии антитела или его антигенсвязывающего фрагмента согласно настоящему изобретению. В таких вариантах осуществления клетки могут быть стабильно трансфицированы вектором, как описано в настоящем документе. Альтернативно, клетки могут быть временно трансфицированы вектором в соответствии с настоящим изобретением, кодирующим антитело или антигенсвязывающий фрагмент, как описано в настоящем документе. В любом из раскрытых в данном документе вариантов осуществления полинуклеотид может быть гетерологичным клетке-хозяину.
Соответственно, настоящее изобретение также относится к рекомбинантным клеткам-хозяевам, которые гетерологично экспрессируют антитело или антигенсвязывающий фрагмент по настоящему изобретению. Например, клетка может быть разновидностью, которая отличается от разновидности, из которой было полностью или частично получено антитело (например, клетки CHO, экспрессирующие человеческое антитело или сконструированное человеческое антитело). В некоторых вариантах осуществления тип клетки клетки-хозяина в природе не экспрессирует антитело или антигенсвязывающий фрагмент. Кроме того, клетка-хозяин может вносить посттрансляционную модификацию (PTM; например, глизоцилирование или фукозилирование) антителу или антигенсвязывающему фрагменту, которое не присутствует в нативном состоянии антитела или антигенсвязывающего фрагмента (или в нативном состоянии исходного антитела, из которого было сконструировано или получено антитело или антигенсвязывающий фрагмент). Такой PTM может привести к функциональной разнице (например, пониженной иммуногенности). Соответственно, антитело или антигенсвязывающий фрагмент согласно настоящему изобретению, продуцируемые клеткой-хозяином, как описано в настоящем документе, могут содержать одну или более посттрансляционных модификаций, которые отличаются от антитела (или исходного антитела) в его нативном состоянии (например, антитело человека, продуцируемое клеткой CHO, может содержать более посттрансляционную модификацию, которая отличается от антитела при выделении из человека и/или продуцируется нативной В-клеткой человека или плазматической клеткой).
Клетки насекомых, пригодные для экспрессии связывающего белка по настоящему изобретению, известны в данной области техники и включают, например, клетки Spodoptera frugipera Sf9, клетки Trichoplusia ni BTI-TN5B1-4 и клетки Spodoptera frugipera SfSWT01“Mimic™”. См., например, Palmberger и др.., J. Biotechnol. 153(3-4):160-166 (2011). Выявлены многочисленные штаммы бакуловирусов, которые можно использовать в комбинации с клетками насекомых, особенно для трансфекции клеток Spodoptera frugiperda.
Эукариотические микроорганизмы, такие как нитчатые грибы или дрожжи, также являются подходящими хозяевами для клонирования или экспрессии векторов, кодирующих белок, и включают грибки и штаммы дрожжей с «гуманизированными» путями гликозилирования, что приводит к получению антитела с частично или полностью человеческим профилем гликозилирования. См . Gerngross, Nat. Biotech.22:1409-1414 (2004); Li et al., Nat. Biotech.24:210-215 (2006).
Растительные клетки также могут быть использованы в качестве хозяев для экспрессии связывающего белка согласно настоящему изобретению. Например, технология PLANTIBODIES™ (описана, например, в патенте США № 5,959,177; 6,040,498; 6,420,548; 7,125,978; и 6,417,429) используют трансгенные растения для получения антител.
В некоторых вариантах осуществления клетка-хозяин включает клетку млекопитающего. В частных вариантах осуществления клетка-хозяин представляет собой клетку СНО, клетку HEK293, клетку PER.C6, клетку Y0, клетку Sp2/0, клетку NS0, клетку печени человека, клетку миеломы или клетку гибридомы.
В родственном аспекте настоящее изобретение относится к способам получения антитела или антигенсвязывающего фрагмента, где способы включают культивирование клетки-хозяина по настоящему изобретению в условиях и в течение времени, достаточных для получения антитела или антигенсвязывающего фрагмента. Способы, пригодные для выделения и очистки рекомбинантно полученных антител, например, могут включать получение супернатантов из подходящих систем клетки-хозяина/вектора, которые секретируют рекомбинантное антитело в культуральную среду, а затем концентрирование среды с использованием коммерчески доступного фильтра. После концентрирования концентрат может быть нанесен на одну подходящую матрицу очистки или на ряд подходящих матриц, таких как аффинная матрица или ионообменная смола. Для дополнительной очистки рекомбинантного полипептида можно использовать одну или более стадий обращенно-фазовой HPLC (высокоэффективная жидкостная хроматография). Эти способы очистки также могут быть использованы при выделении иммуногена из его природной среды. Способы крупномасштабной продукции одного или более выделенных/рекомбинантных антител, описанных в настоящем документе, включают периодическую культуру клеток, за которой наблюдали и контролировали для поддержания соответствующих условий культивирования. Очистку растворимых антител можно проводить в соответствии со способами, описанными в настоящем документе и известными в данной области техники, и которые соответствуют законам и рекомендациям отечественных и зарубежных регулирующих органов.
Композиции
В настоящем документе также предложены композиции, которые содержат любое одно или более из описанных в настоящем документе антител, антигенсвязывающих фрагментов, полинуклеотидов, векторов или клеток-хозяев, по отдельности или в любой комбинации, и могут дополнительно содержать фармацевтически приемлемый носитель, вспомогательное вещество или разбавитель. Носители, эксципиенты и разбавители более подробно обсуждаются в данном документе.
В некоторых вариантах осуществления композиция содержит множество антител и/или антигенсвязывающих фрагментов по настоящему изобретению, где одно или более антител или антигенсвязывающих фрагментов не содержит остаток лизина на С-конце тяжелой цепи, CH1-CH3 или полипептида Fc, и где одно или более антител или антигенсвязывающих фрагментов содержит остаток лизина на С-конце тяжелой цепи, CH1-CH3 или полипептида Fc. В некоторых вариантах осуществления изобретения композиция содержит антитело или его антигенсвязывающий фрагмент, которые содержат аминокислотную последовательность, представленную в SEQ ID NO:173, SEQ ID NO:175, SEQ ID NO:265 или SEQ ID NO:266.
В некоторых вариантах осуществления композиция содержит два или более различных антитела или антигенсвязывающие фрагменты в соответствии с настоящим изобретением. В некоторых вариантах осуществления антитела или антигенсвязывающие фрагменты для применения в комбинации, каждая независимо, имеют одну или более из следующих характеристик: нейтрализуют встречающиеся в природе варианты SARS-CoV-2; не конкурируют друг с другом за связывание со спайк-белком; связывают различные эпитопы спайк-белка; имеют сниженное образование устойчивости к SARS-CoV-2; в комбинации имеют сниженное образование устойчивости к SARS-CoV-2; эффективно нейтрализуют живой вирус SARS-CoV-2; проявляют аддитивные или синергетические эффекты на нейтрализацию живого вируса SARS-CoV-2 при использовании в комбинации; проявляют эффекторные функции; являются защитными в соответствующей модели (моделях) инфекции животного; способны продуцироваться в достаточных количествах для крупномасштабной продукции.
В некоторых вариантах осуществления композиция содержит первое антитело или антигенсвязывающий фрагмент, содержащее VH, содержащий или состоящий из аминокислотной последовательности, представленной в SEQ ID NO: 79, и VL, содержащий или состоящий из аминокислотной последовательности, представленной в SEQ ID NO: 83; и второе антитело или антигенсвязывающий фрагмент, содержащий, VH, содержащий или состоящий из аминокислотной последовательности, представленной в SEQ ID NO: 105, и VL, содержащий или состоящий из аминокислотной последовательности, представленной в SEQ ID NO: 168. В некоторых вариантах осуществления композиция содержит первое антитело или антигенсвязывающий фрагмент, содержащий вариабельный домен тяжелой цепи (VH), содержащий CDRH1, CDRH2 и CDRH3, и вариабельный домен легкой цепи (VL), содержащий CDRL1, CDRL2 и CDRL3, где CDRH1, CDRH2 и CDRH3 содержат или состоят из аминокислотных последовательностей, представленных в SEQ ID NO: 80-82, соответственно, и CDRL1, CDRL2 и CDRL3 содержат или состоят из аминокислотных последовательностей, представленных в SEQ ID NO: 84-86, соответственно, и второе антитело или антигенсвязывающий фрагмент, содержащий вариабельный домен тяжелой цепи (VH), содержащий CDRH1, CDRH2 и CDRH3, и вариабельный домен легкой цепи (VL), содержащий CDRL1, CDRL2 и CDRL3, где CDRH1, CDRH2 и CDRH3 содержат или состоят из аминокислотных последовательностей, представленных в SEQ ID NO: 106-108, соответственно, и CDRL1 и CDRL3, или состоят из аминокислотных последовательностей, представленных в SEQ ID NO: 169-171, соответственно. В дополнительных вариантах осуществлений композиция способна нейтрализовать инфекцию SARS-CoV-2 или вирус, псевдотипированный SARS-CoV-2, с IC50 от около 0,07 до около 0,08 мкг/мл. В некоторых вариантах осуществления композиция содержит первое антитело или антигенсвязывающий фрагмент, содержащее VH, содержащий или состоящий из аминокислотной последовательности, представленной в SEQ ID NO: 178, и VL, содержащий или состоящий из аминокислотной последовательности, представленной в SEQ ID NO: 182 или SEQ ID NO: 190; и второе антитело или антигенсвязывающий фрагмент, содержащее VH, содержащий или состоящий из аминокислотной последовательности, представленной в SEQ ID NO: 105, и VL, содержащий, состоящий из аминокислотной последовательности, представленной в SEQ ID NO: 168. В некоторых вариантах осуществления композиция содержит первое антитело или антигенсвязывающий фрагмент, содержащее вариабельный домен тяжелой цепи (VH), содержащий CDRH1, CDRH2 и CDRH3, и вариабельный домен легкой цепи (VL), содержащий CDRL1, CDRL2 и CDRL3, где CDRH1, CDRH2 и CDRH3 содержат или состоят из аминокислотных последовательностей, представленных в SEQ ID NO: 179-181, соответственно, и CDRL1, CDRL2 и CDRL3 содержат или состоят из аминокислотных последовательностей, представленных в SEQ ID NO: 183-185, соответственно, и второе антитело или антигенсвязывающий фрагмент, содержащее вариабельный домен тяжелой цепи (VH), содержащий CDRH1, CDRH2 и CDRH3, и вариабельный домен легкой цепи (VL), содержащий CDRL1, CDRL2 и CDRL3, где CDRH1, CDRH2 и CDRH3 содержат или состоят из аминокислотных последовательностей, представленных в SEQ ID NO: 106-108, соответственно, и CDRL1, CDRL2 и CDRL3 содержат аминокислотные последовательности, представленные в SEQ ID NO: 169-171, соответственно.
В некоторых вариантах осуществления композиция содержит первый вектор, содержащий первую плазмиду, и второй вектор, содержащий вторую плазмиду, где первая плазмида содержит полинуклеотид, кодирующий тяжелую цепь, VH или VH+CH, а вторая плазмида содержит полинуклеотид, кодирующий родственную легкую цепь, VL или VL+CL антитела или его антигенсвязывающего фрагмента. В некоторых вариантах осуществления композиция содержит полинуклеотид (например, мРНК), связанный с подходящим средством доставки или носителем. Примерные носители или носители для введения субъекту-человеку включают липид или липидное средство доставки, такой как липосома, твердая липидная наночастица, маслянистая суспензия, субмикронная липидная эмульсия, липидный микропузырь, обратная липидная мицелла, кохлеарная липосома, липидная микротрубочка, липидный микроцилиндр или липидная наночастица (LNP) или наноразмерная платформа (см., например, Li и др. Wilery Interdiscip Rev. Nanomed Nanobiotechnol. 11(2):e1530 (2019)). Принципы, реагенты и методики конструирования подходящей мРНК и составления мРНК-LNP и доставки их описаны, например, в Pardi и др. (J Control Release 217345-351(2015)); Thess и др.. (Mol Ther 23: 1456-1464 (2015)); Thran и др. (EMBO Mol Med 9(10):1434-1448 (2017); Kose и др. Sci. Immunol. 4 eaaw6647 (2019); и Sabnis и др. (Mol. Ther. 26: 1509-1519 (2018)), которые включают кэппинг, оптимизацию кодонов, нуклеозидную модификацию, очистку мРНК, включение мРНК в стабильные липидные наночастицы (например, ионизируемый катионный липид/фосфатидилхолин/холестерин/PEG-липид (PEG - полиэтиленгликоль); ионизируемый липид:дистеароил PC:холестерин:полиэтиленгликолевый липид (PC - фосфатидилхолин)) и подкожное, внутримышечное, внутрикожное, внутривенное, внутрибрюшинное и интратрахеальное введение, включены в настоящий документ посредством ссылки.
Способы и применения
В настоящем документе также предложены способы применения антитела или антигенсвязывающего фрагмента, нуклеиновой кислоты, вектора, клетки или композиции по настоящему изобретению при диагностике SARS-CoV-2 (например, у субъекта-человека или в образце, полученном от субъекта-человека).
Способы диагностики (например, in vitro, ex vivo) могут включать приведение в контакт антитела, фрагмента антитела (например, антигенсвязывающего фрагмента) с образцом. Такие образцы могут быть выделены из субъекта, например, выделенный образец ткани, взятый, например, из носовых ходов, полостей пазух, слюнных желез, легких, печени, поджелудочной железы, почек, уха, глаза, плаценты, пищеварительного тракта, сердца, яичников, гипофиза, надпочечников, щитовидной железы, мозга, кожи или крови. Способы диагностики могут также включать обнаружение комплекса антиген/антитело, в частности, после приведения в контакт антитела или фрагмента антитела с образцом. Такой этап обнаружения может быть выполнен в ламинарном шкафе, то есть без какого-либо приведения в контакт с организмом человека или животного. Примеры способов обнаружения хорошо известны специалисту в данной области техники и включают, например, ELISA (твердофазный иммуноферментный анализ), включая прямой, непрямой и многослойный ELISA.
В настоящем документе также предложены способы лечения субъекта с использованием антитела или антигенсвязывающего фрагмента по настоящему изобретению, или композиции, содержащей их, причем субъект имеет, как полагают, или подвержен риску инфицирования SARS-CoV-2. "Обработка", "лечение" или "облегчение" относится к медицинскому лечению заболевания, расстройства или состояния субъекта (например, человека или млекопитающего, не являющегося человеком, такого как примат, лошадь, кошка, собака, коза, мышь или крыса). Как правило, подходящую дозу или схему лечения, включающую антитело или композицию по настоящему изобретению, вводили в количестве, достаточном для получения терапевтического или профилактического эффекта. Терапевтический или профилактический/превентивный эффект включает улучшение клинического исхода; уменьшение или облегчение симптомов, связанных с заболеванием; уменьшение частоты возникновения симптомов; улучшение качества жизни; более длительный безрецидивный статус; уменьшение степени заболевания, стабилизацию состояния заболевания; задержку или предотвращение прогрессирования заболевания; ремиссию; выживаемость; продолжительную выживаемость; или любую их комбинацию. В некоторых вариантах осуществления терапевтический, профилактический/превентивный эффект включает уменьшение или предотвращение госпитализации прир лечении инфекции SARS-CoV-2 (т.е. статистически значимым образом). В некоторых вариантах осуществления терапевтический или профилактическоий/превентивный эффект включает сокращение продолжительности госпитализации при лечении инфекции SARS-CoV-2 (т.е. статистически значимым образом). В некоторых вариантах осуществления терапевтический или профилактический/превентивный эффект включает уменьшение или отмену потребности в респираторном вмешательстве, таком как интубация и/или применение респираторного устройства. В некоторых вариантах осуществления изобретения терапевтический или профилактический/превентивный эффект включает реверсию патологии поздней стадии заболевания и/или снижение смертности.
«Терапевтически эффективное количество» или «эффективное количество» антитела, антигенсвязывающего фрагмента, полинуклеотида, вектора, клетки-хозяина или композиции согласно настоящему изобретению относится к количеству композиции или молекулы, достаточному для получения терапевтического эффекта, включая улучшение клинического исхода; уменьшение или облегчение симптомов, связанных с заболеванием; снижение частоты возникновения симптомов; улучшение качества жизни; более длительный безрецидивный статус; уменьшение степени заболевания, стабилизация состояния заболевания; задержку прогрессирования заболевания; ремиссию; выживаемость; или продолжительное выживание статистически значимым образом. Когда речь идет об отдельном активном ингредиенте, вводимом отдельно, терапевтически эффективное количество относится к эффектам этого ингредиента или клетки, экспрессирующей этот ингредиент отдельно. Когда речь идет о комбинации, терапевтически эффективное количество относится к объединенным количествам активных ингредиентов или комбинированного вспомогательного активного ингредиента с клеткой, экспрессирующей активный ингредиент, что приводит к терапевтическому эффекту, независимо от того, вводится ли оно серийно, последовательно или одновременно. Комбинация может содержать, например, два различных антитела, которые специфически связывают антиген SARS-CoV-2, который в некоторых вариантах осуществления может представлять собой один и тот же или другой антиген коронавируса Wuhan, и/или может содержать одинаковые или разные эпитопы.
Соответственно, в некоторых вариантах осуществления предложены способы лечения инфекции SARS-CoV-2 у субъекта, где способы включают введение указанному субъекту эффективного количества антитела, антигенсвязывающего фрагмента, полинуклеотида, вектора, клетки-хозяина или композиции, как описано в настоящем документе.
Субъекты, которые могут быть подвергнуты лечению согласно настоящему изобретению, являются, в целом, субъектами людьми и другими приматами, такими как мартышки и обезьяны, для целей ветеринарной медицины. Другие модели организмов, такие как мыши и крысы, также могут быть подвергнуться лечению в соответствии с настоящим изобретением. В любом из вышеупомянутых вариантов осуществления субъект может быть субъектом человеком. Субъекты могут быть мужского или женского пола и могут быть любого подходящего возраста, включая младенцев, несовершеннолетних, подростков, взрослых и пожилых субъектов.
Считается, что ряд критериев способствует высокому риску тяжелых симптомов или смерти, связанных с инфекцией коронавируса SARS CoV-2. Они включают, но не ограничиваются этим, возраст, род занятий, общее состояние здоровья, ранее существовавшие состояния здоровья и привычки образа жизни. В некоторых вариантах осуществления субъект, получающий лечение в соответствии с настоящим изобретением, содержит один или более факторов риска.
В некоторых вариантах осуществления субъект человек, получающий лечение в соответствии с настоящим изобретением, представляет собой младенца, ребенка, подростка, взрослого среднего возраста или пожилого человека. В некоторых вариантах осуществления субъект человек, получающий лечение в соответствии с настоящим изобретением, моложе 1 года, или ему от 1 до 5 лет, или ему от 5 до 125 лет (например, 5, 10, 15, 20, 25, 30, 35, 40, 45, 50, 55, 60, 65, 70, 75, 80, 85, 90, 95, 100, 105, 110, 115 или 125 лет, включая любой и все возрасты в списке или между указанными возрастами). В некоторых вариантах осуществления возраст субъекта-человека, получающего лечение в соответствии с настоящим изобретением, составляет 0-19 лет, 20-44 года, 45-54 года, 55-64 года, 65-74 года, 75-84 года или 85 лет или старше. Особому риску, как полагают, подвергаются лица среднего и особенно пожилого возраста. В частных вариантах осуществления возраст субъекта-человека составляет 45-54 года, 55-64 года, 65-74 года, 75-84 года или 85 лет или старше. В некоторых вариантах осуществления субъектом человеком является мужчина. В некоторых вариантах осуществления субъект человек является женщина.
В некоторых вариантах осуществления субъект человек, получающий лечение в соответствии с настоящим изобретением, является резидентом дома престарелых или учреждения долгосрочного ухода, является работником хосписа, является медицинским работником или медицинским работником, является сотрудником скорой помощи, является членом семьи или другим человеком, находящемся в близком контакте с субъектом, у которого диагностирована или подозревается инфекция SARS-CoV-2, имеет избыточный вес или клинически ожирение, курит или курил, имеет или имел хроническое обструктивное заболевание легких (COPD), является астматиком (например, имеет умеренную или тяжелую астму), имеет аутоиммунное заболевание или состояние (например, диабет) и/или имеет ослабленную или истощенную иммунную систему (например, в связи со СПИД/ВИЧ-инфекцией (СПИД - синдром инфекции иммунодефицита, ВИЧ - вирус иммунодефицита человека), раком, таким как рак крови, лимфодеплетирующей терапией, такой как химиотерапия, трансплантацей костного мозга или органа, или генетическим иммунным состояние), имеет хроническое заболевание печени, имеет сердечно-сосудистое заболевание, заболевание легких или дефект сердца, работает или иным образом проводит время в непосредственной близости с другими, например, на фабрике, в центре доставки, в больнице и т.п.
В некоторых вариантах осуществления изобретения субъект, получающий лечение в соответствии с настоящим изобретением, получил вакцину против SARS-CoV-2, и вакцина была признана неэффективной, например, в результате поствакцинной инфекции или симптомов у субъекта, в результате клинического диагноза или научных или нормативных критериев.
В некоторых вариантах осуществления лечение осуществляли в качестве профилактики после контакта. В некоторых вариантах осуществления лечение осуществляли субъекту с заболеванием легкой или умеренной степени, который может находиться в амбулаторных условиях. В некоторых вариантах осуществлениях лечение осуществляли субъекту с заболеванием средней или тяжелой степени, например, требующим госпитализации.
Таким образом, типичные пути введения описанных в настоящем документе композиций включают, без ограничения, пероральный, местный, трансдермальный, ингаляционный, парентеральный, сублингвальный, буккальный, ректальный, вагинальный и интраназальный. Термин "парентеральный", как используется в настоящем документе, включает подкожные инъекции, внутривенные, внутримышечные, внутригрудинный инъекции или методики инфузий. В некоторых вариантах осуществления введение включает введение путем, выбранным из перорального, внутривенного, парентерального, внутрижелудочного, внутриплеврального, внутрилегочного, внутриректального, интрадермального, внутрибрюшинного, внутриопухолевого, подкожного, местного, трансдермального, внутричерепного, интратекального, интраназального и внутримышечного введения. В частных вариантах осуществления способ включает пероральное введение субъекту антитела, антигенсвязывающего фрагмента, полинуклеотида, вектора, клетки-хозяина или композиции.
Фармацевтические композиции в соответствии с некоторыми вариантами осуществления настоящего изобретения составлены таким образом, чтобы обеспечить биодоступность содержащихся в ней активных ингредиентов при введении композиции пациенту. Композиции, которые будут вводит субъекту или пациенту, могут иметь форму одной или более единиц дозирования, где, например, таблетка может представлять собой единичную единицу дозирования, и контейнер описанного в настоящем документе антитела или антигенсвязывающего фрагмента в аэрозольной форме может содержать множество дозированных единиц. Реальные способы получения таких дозированных форм известны или очевидны специалистам в данной области; например, см. Remington: The Science and Practice of Pharmacy, 20th Edition (Philadelphia College of Pharmacy and Science, 2000). Композиция, подлежащая введению, в любом случае будет содержать эффективное количество антитела или антигенсвязывающего фрагмента, полинуклеотида, вектора, клетки-хозяина или композиции по настоящему изобретению для лечения заболевания или состояния, представляющего интерес, в соответствии с настоящим изобретением.
Композиция может быть в форме твердого вещества или жидкости. В некоторых вариантах осуществления носитель (носители) являются твердыми частицами, так что композиции находятся, например, в форме таблетки или порошка. Носитель (носители) может быть жидким (жидкими), так что композиции представляют собой, например, масло для полости рта, инъекционную жидкость или аэрозоль, который можно применять, например, при ингаляционном введении. Когда фармацевтическая композиция предназначена для перорального введения, она предпочтительно находится либо в твердой, либо в жидкой форме, где полутвердая, полужидкая, суспензионная и гелевая формы включены в формы, рассматриваемые в настоящем документе как твердые или жидкие.
В качестве твердой композиции для перорального введения фармацевтическая композиция может быть составлена в виде порошка, гранулы, прессованной таблетки, пилюли, капсулы, жевательной резинки, облатки или т. п. Такая твердая композиция обычно содержит один или более инертных разбавителей или съедобных носителей. Кроме того, могут присутствовать одно или более из следующих веществ: связующие вещества, такие как карбоксиметилцеллюлоза, этилцеллюлоза, микрокристаллическая целлюлоза, трагакантовая камедь или желатин; вспомогательные вещества, такие как крахмал, лактоза или декстрины, дезинтегрирующие агенты, такие как альгиновая кислота, альгинат натрия, примогель, кукурузный крахмал и т. п.; смазывающие вещества, такие как стеарат магния или стеротекс; скользящие агенты, такие как коллоидный диоксид кремния; подсластители, такие как сахароза или сахарин; ароматизатор, такой как перечная мята, метилсалицилат или апельсиновый ароматизатор; и краситель. Когда композиция находится в форме капсулы, например, желатиновой капсулы, она может содержать, помимо веществ вышеуказанного типа, жидкий носитель, такой как полиэтиленгликоль или масло.
Композиция может быть в форме жидкости, например, эликсира, сиропа, раствора, эмульсии или суспензии. Жидкость может быть предназначена для перорального введения или для доставки путем инъекции, в качестве двух примеров. Если они предназначены для перорального введения, предпочтительные композиции содержат в дополнение к настоящим соединениям один или более подсластителей, консервантов, красящих веществ/красителей и усилителей вкуса. В композицию, предназначенную для введения путем инъекции могут быть включены одно или более из поверхностно-активного вещества, консерванта, смачивающего агента, диспергирующего агента, суспендирующего агента, буфера, стабилизатора и изотонического агента.
Жидкие фармацевтические композиции, будь то растворы, суспензии или другие подобные формы, могут включать один или несколько из следующих адъювантов: стерильные разбавители, такие как вода для инъекций, физиологический раствор, предпочтительно физиологический раствор, раствор Рингера, изотонический хлорид натрия, жирные масла, такие как синтетические моно- или диглицериды, которые могут служить растворителем или суспендирующей средой, полиэтиленгликоли, глицерин, пропиленгликоль или другие растворители; антибактериальные агенты, такие как бензиловый спирт или метилпарабен; антиоксиданты, такие как аскорбиновая кислота или бисульфит натрия; хелатообразующие агенты, такие как этилендиаминтетрауксусная кислота; буферы, такие как ацетаты, цитраты или фосфаты и агенты для достижения тоничности, такие как хлорид натрия или декстроза. Парентеральный препарат может быть включен в ампулы, одноразовые шприцы или флакон из стекло или пластика с несколькими дозами. Физиологический раствор является предпочтительным адъювантом. Инъецируемая фармацевтическая композиция, предпочтительно, является стерильной.
Жидкая композиция, предназначенная для парентерального или перорального введения, должна содержать количество антитела или антигенсвязывающего фрагмента, как описано в настоящем документе, таким образом, чтобы была получена подходящая дозировка. Как правило, это количество составляет по меньшей мере 0,01% от антитела или антигенсвязывающего фрагмента в композиции. Когда это предназначено для перорального введения, это количество может варьироваться от 0,1 до около 70% от массы композиции. Некоторые пероральные фармацевтические композиции содержат от около 4% до около 75% антитела или антигенсвязывающего фрагмента. В некоторых вариантах осуществления фармацевтические композиции и препараты согласно настоящему изобретению получали таким образом, что парентеральная единица дозирования содержит от 0,01 до 10% по массе антитела или антигенсвязывающего фрагмента перед разведением.
Композиция может быть предназначена для местного введения, и в этом случае носитель может подходящим образом содержать раствор, эмульсию, мазь или гелевую основу. Основа, например, может содержать одно или более из следующего: петролатум, ланолин, полиэтиленгликоли, пчелиный воск, минеральное масло, разбавители, такие как вода и спирт, и эмульгаторы и стабилизаторы. Загустители могут присутствовать в композиции для местного введения. Если композиция предназначена для трансдермального введения, она может включать трансдермальный пластырь или устройство для ионофореза. Фармацевтическая композиция может быть предназначена для ректального введения, в форме, например, суппозитория, который будет растворяться в прямой кишке и высвобождать лекарственное средство. Композиция для ректального введения может содержать маслянистую основу в качестве подходящего не раздражающего вспомогательного вещества. Такие основы включают, без ограничения, ланолин, масло какао и полиэтиленгликоль.
Композиция может содержать различные вещеста, которые модифицируют физическую форму твердой или жидкой единицы дозирования. Например, композиция может включать материалы, которые образуют оболочку покрытия вокруг активных ингредиентов. Вещества, которые образуют оболочку покрытия, обычно являются инертными и могут быть выбраны, например, из сахара, шеллака и других энтеросолюбильных покрывающих агентов. Альтернативно активные ингредиенты могут быть заключены в желатиновую капсулу. Композиция в твердой или жидкой форме может содержать агент, который связывается с антителом или антигенсвязывающим фрагментом согласно настоящему описанию и тем самым способствует доставке соединения. Подходящие агенты, которые могут действовать в этом качестве, включают моноклональные или поликлональные антитела, один или более белков или липосому. Композиция может состоять по существу из единиц дозирования, которые можно вводить в виде аэрозоля. Термин "аэрозоль" используется для обозначения различных систем, начиная с систем коллоидной природы и кончая системами, состоящими из упаковок под давлением. Доставка может осуществляться сжиженным или сжатым газом или с помощью подходящей насосной системы, которая дозирует активные ингредиенты. Аэрозоли могут доставляться в однофазных, двухфазных или трехфазных системах для доставки активного (активныых) ингредиента (ингридиентов). Доставка аэрозоля включает необходимый контейнер, активаторы, клапаны, субконтейнеры и т. п., которые вместе могут образовывать набор. Специалист в данной области техники без излишних экспериментов может определить предпочтительные аэрозоли.
Следует понимать, что композиции по настоящему изобретению также охватывают молекулы-носители для полинуклеотидов, как описано в настоящем документе (например, липидные наночастицы, платформы доставки в наноразмерном масштабе и т. п.).
Фармацевтические композиции могут быть получены с помощью методики, хорошо известной в данной области техники. Например, композиция, предназначенная для введения путем инъекции, может быть получена путем комбинирования композиции, которая содержит антитело, его антигенсвязывающий фрагмент или конъюгат антитела, как описано в настоящем документе, и, необязательно, одну или более солей, буферов и/или стабилизаторов, со стерильной дистиллированной водой с образованием раствора. Поверхностно-активное вещество может быть добавлено для облегчения образования гомогенного раствора или суспензии. Поверхностно-активные вещества представляют собой соединения, которые нековалентно взаимодействуют с пептидной композицией таким образом, чтобы облегчить растворение или гомогенную суспензию антитела или его антигенсвязывающего фрагмента в водной системе доставки.
В целом, подходящая доза и схема лечения обеспечивают композицию (композиции) в количестве, достаточном для обеспечения терапевтического и/или профилактического эффекта (такого, как описано в настоящем документе, включая улучшенный клинический исход (например, снижение частоты, продолжительности или тяжести диареи или связанного с ней обезвоживания, или воспаления, или более длительной безрецидивной и/или общей выживаемости, или уменьшение тяжести симптомов). Для профилактического применения доза должна быть достаточной для предотвращения, задержки возникновения или уменьшения тяжести заболевания, связанного с заболеванием или расстройством. Профилактический эффект композиций, вводимых в соответствии со способами, описанными в настоящем документе, может быть определена путем выполнения доклинических (включая исследования на животных in vitro и in vivo) и клинических исследований и анализа данных, полученных из них, с помощью соответствующих статистических, биологических и клинических способов и методик, все из которых могут быть легко осуществлены специалистом в данной области техники.
Композиции вводили в эффективном количестве (например, для лечения инфекции SARS-CoV-2), которое будет варьироваться в зависимости от множества факторов, включая активность конкретного применяемого соединения; метаболическую стабильность и продолжительность действия соединения; возраст, массу тела, общее состояние здоровья, пол и диету субъекта; режим и время введения; скорость выведения; комбинацию лекарственного средства; тяжесть конкретного расстройства или состояния; и субъект, проходящий терапию. В некоторых вариантах осуществления настоящего изобретения при последующем введении лекарственных средств в соответствии с составами и способами согласно настоящему изобретению у субъектов будет наблюдаться снижение одного или более симптомов, связанных с заболеванием или расстройством, подлежащим лечению, на от около 10% до около 99% по сравнению с субъектами, получающими плацебо, или другими подходящими контрольными субъектами.
Как правило, терапевтически эффективная суточная доза антитела или антигенсвязывающего фрагмента составляет (для млекопитающего весом 70 кг) от около 0,001 мг/кг (т.е.0,07 мг) до около 100 мг/кг (т.е. 7,0 г); предпочтительно терапевтически эффективная доза составляет (для млекопитающего весом 70 кг) от около 0,01 мг/кг (т.е.0,7 мг) до около 50 мг/кг (т.е.3,5 г); более предпочтительно терапевтически эффективная доза составляет (для млекопитающего весом 70 кг) от около 1 мг/кг (т.е.70 мг) до около 25 мг/кг (т.е.1,75 г). Для полинуклеотидов, векторов, клеток-хозяев и связанных композиций по настоящему изобретению терапевтически эффективная доза может отличаться от дозы для антитела или антигенсвязывающего фрагмента.
В некоторых вариантах осуществления способ включает введение антитела, антигенсвязывающего фрагмента, полинуклеотида, вектора, клетки-хозяина или композиции субъекту в количестве 2, 3, 4, 5, 6, 7, 8, 9, 10 раз или более.
В некоторых вариантах осуществления способ включает введение антитела, антигенсвязывающего фрагмента или композиции субъекту множество раз, где второе или последовательное введение осуществляли через около 6, около 7, около 8, около 9, около 10, около 11, около 12, около 24, около 48, около 74, около 96 часов или более после первого или предыдущего введения, соответственно.
В некоторых вариантах осуществления способ включает введение антитела, антигенсвязывающего фрагмента, полинуклеотида, вектора, клетки-хозяина или композиции по меньшей мере один раз до инфицирования субъекта SARS-CoV-2.
Композиции, содержащие антитело, антигенсвязывающий фрагмент, полинуклеотид, вектор, клетку-хозяина или композицию по настоящему изобретению, также можно вводить одновременно, до или после введения одного или более других терапевтических средств. Такая комбинированная терапия может включать введение одной фармацевтической лекарственной формы, которая содержит соединение по изобретению и один или более дополнительных активных агентов, а также введение композиций, содержащих антитело или антигенсвязывающий фрагмент по изобретению и каждый активный агент в своей отдельной лекарственной форме. Например, антитело или его антигенсвязывающий фрагмент, как описано в настоящем документе, и другой активный агент можно вводить пациенту вместе в одной пероральной лекарственной композиции, такой как таблетка или капсула, или каждый агент вводили в отдельных пероральных лекарственных формах. Аналогичным образом, антитело или антигенсвязывающий фрагмент, как описано в настоящем документе, и другой активный агент могут быть введены субъекту вместе в одной парентеральной лекарственной композиции, такой как физиологический раствор или другой физиологически приемлемый раствор, или каждый агент может быть введен в отдельных парентеральных лекарственных формах. В тех случаях, когда используются отдельные лекарственные формы, композиции, содержащие антитело или антигенсвязывающий фрагмент и один или более дополнительных активных агентов, могут быть введены по существу в одно и то же время, то есть одновременно, или в разное время, то есть последовательно и в любом порядке; предполагается, что комбинированная терапия включает все эти схемы.
В некоторых вариантах осуществления настоящего изобретения предлагается комбинированная терапия, которая содержит одно или более антител к SARS-CoV-2 (или одну или более нуклеиновых кислот, клеток-хозяев, векторов или композиций) по настоящему изобретению и один или более противовоспалительных средств и/или один или более противовирусных средств. В конкретных вариантах осуществления один или более противовоспалительных средств включает кортикостероид, такой как, например, дексаметазон, преднизон или т. п. В некоторых вариантах осуществления один или более противовоспалительных средств включает антагонист цитокина, такой как, например, антитело, которое связывается с IL6 (IL -интерлейкин) (таким как силтуксимаб), или с IL-6R (рецептор IL-6) (таким как тоцилизумаб), или с IL-1β, IL-7, IL-8, IL-9, IL-10, FGF (фактор роста фибробластов), G-CSF (колониестимулирующий фактор гранулоцитов), GM-CSF (гранулоцитарно-макрофагальный колониестимулирующий фактор), IFN-γ (интерферон γ), IP-10 (интерферон-γ-индуцируемый белок 10), MCP-1 (моноцитарный хемоатрактантный белок), MIP-1A (MIP -макрофагальный белок воспаления), MIP1-B, PDGR (тромбоцитарный фактор роста), TNF-α (фактор некроза опухоли α) или VEGF (фактор роста эндотелия сосудов). В некоторых вариантах осуществления применяли противовоспалительные средства, такие как руксолитиниб и/или анакинра. В некоторых вариантах осуществления один или более противовирусных средств содержат нуклеотидные аналоги или пролекарства нуклетидных аналогов, такие как, например, ремдесивир, софосбувир, ацикловир и зидовудин. В частных вариантах осуществления противовирусное средство включает лопинавир, ритонавир, фавипиравир или любую их комбинацию. В некоторых вариантах осуществления комбинированная терапия включает леронлимаб. Противовоспалительные средства для применения в комбинированной терапии по настоящему изобретению также включают нестероидные противовоспалительные средства (NSAID). Следует понимать, что в такой комбинированной терапии одно или более антител (или одна или более нуклеиновых кислот, клеток-хозяев, векторов или композиций) и одно или более противовоспалительных средств и/или одно или более противовирусных средств могут быть введены в любом порядке и в любой последовательности или вместе.
В некоторых вариантах осуществления антитело (или одна или более нуклеиновых кислот, клеток-хозяев, векторов или композиций) вводили субъекту, который ранее получал один или более противовоспалительных средств и/или один или более противовирусных средств. В некоторых вариантах осуществления один или более противовоспалительных средств и/или один или более противовирусных средств вводили субъекту, которому ранее вводили антитело (или одну или более нуклеиновых кислот, клеток-хозяев, векторов или композиций).
В некоторых вариантах осуществления настоящего изобретения предлагается комбинированная терапия, которая содержит два или более антител против SARS-CoV-2 по настоящему изобретению. Способ может включать введение первого антитела субъекту, который получил второе антитело, или может включать введение двух или более антител вместе. Например, в частных вариантах осуществления предложен способ, который включает введение субъекту (а) первого антитела или антигенсвязывающего фрагмента, когда субъект получил второе антитело или антигенсвязывающий фрагмент; (b) второго антитела или антигенсвязывающего фрагмента, когда субъект получил первое антитело или антигенсвязывающий фрагмент; или (с) первого антитела или антигенсвязывающего фрагмента и второго антитела или антигенсвязывающего фрагмента.
В связанном аспекте предлагаются применения описанных в настоящем документе антител, антигенсвязывающих фрагментов, векторов, клеток-хозяев и композиций.
В некоторых вариантах осуществления предлагается антитело, антигенсвязывающий фрагмент, полинуклеотид, вектор, клетка-хозяин или композиция для применения в способе лечения инфекции SARS-CoV-2 у субъекта.
В некоторых вариантах осуществления данного изобретения предлагается антитело, антигенсвязывающий фрагмент или композиция для применения в способе изготовления или получения лекарственного средства для лечения инфекции SARS-CoV-2 у субъекта.
Настоящее изобретение также относится к следующим вариантам осуществления.
Вариант осуществления 1. Антитело или его антигенсвязывающий фрагмент, содержащее вариабельный домен тяжелой цепи (VH), содержащий CDRH1 (область 1, определяющая комплементарность тяжелой цепи), CDRH2 и CDRH3, и вариабельный домен легкой цепи (VL), содержащий CDRL1 (область 1, определяющая комплементарность легкой цепи), CDRL2 и CDRL3, где:
(i) CDRH1 содержит или состоит из аминокислотной последовательности, представленной в любой из SEQ ID NO:106, 2, 56, 64, 80, 88, 96, 156, 179, 195 или 240, или ее варианта последовательности, содержащего одну, две или три аминокислотные замены, одна или более из которых необязательно представляет собой консервативную замену и/или является заменой аминокислоты, кодируемой зародышевой линией;
(ii) CDRH2 содержит или состоит из аминокислотной последовательности, представленной в любой из SEQ ID NO:121, 3, 16-22, 57, 65, 81, 89, 97, 107, 122-126, 157, 180, 197, 199 или 241, или ее варианта последовательности, содержащего одну, две или три аминокислотные замены, одна или более из которых необязательно представляет собой консервативную замену и/или является заменой аминокислоты, кодируемой зародышевой линией;
(iii) CDRH3 содержит или состоит из аминокислотной последовательности, представленной в любой из SEQ ID NO:108, 4, 25, 26, 58, 66, 82, 90, 98, 104, 127, 128, 158, 181, 201, 203 или 242, или ее варианта последовательности, содержащего одну, две или три аминокислотные замены, одна или более из которых необязательно представляет собой консервативную замену и/или является заменой аминокислоты, кодируемой зародышевой линией;
(iv) CDRL1 содержит или состоит из аминокислотной последовательности, представленной в любой из SEQ ID NO:169, 6, 51-54, 60, 68, 73, 74, 84, 92, 100, 110, 160, 183, 235 или 244, или ее варианта последовательности, содержащего одну, две или три аминокислотные замены, одна или более из которых необязательно представляет собой консервативную замену и/или является заменой аминокислоты, кодируемой зародышевой линией;
(v) CDRL2 содержит или состоит из аминокислотной последовательности, представленной в любой из SEQ ID NO:170, 7, 61, 69, 85, 93, 101, 111, 161, 184, 236 или 245, или ее варианта последовательности, содержащего одну, две или три аминокислотные замены, одна или более из которых необязательно представляет собой консервативную замену и/или является заменой аминокислоты, кодируемой зародышевой линией; и/или
(vi) CDRL3 содержит или состоит из аминокислотной последовательности, представленной в любой из SEQ ID NO:171, 8, 62, 70, 77, 78, 86, 94, 102, 112, 151-154, 162, 185, 237 или 246, или ее варианта последовательности, содержащего одну, две или три аминокислотные замены, одна или более из которых необязательно представляет собой консервативную замену и/или является заменой аминокислоты, кодируемой зародышевой линией,
где антитело или антигенсвязывающий фрагмент способно связываться с поверхностным гликопротеином (S) SARS-CoV-2, экспрессируемом на клеточной поверхности клетки-хозяина, на вирионе SARS-CoV-2 или на обоих.
Вариант осуществления 2. Антитело или его антигенсвязывающий фрагмент по варианту осуществления 1, способное нейтрализовать инфекцию SARS-CoV-2 на модели инфекции in vitro и/или на животной модели инфекции in vivo и/или у человека.
Вариант осуществления 3. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 1 или 2, содержащее аминокислотные последовательности CDRH1, CDRH2, CDRH3, CDRL1, CDRL2 и CDRL3, представленные в SEQ ID NO:
(i) 106, 121, 108, 169, 170 и 171, соответственно;
(ii) 2-4 и 6-8 или 235-237, соответственно;
(iii) 2, любой из 16-22, 4 и 6-8 или 235-237, соответственно;
(iv) 2, 3, любой из 25-26 и 6-8 или 235-237, соответственно;
(v) 2-4, 51, 7 или 236 и 8 или 237, соответственно;
(vi) 2-4, 52, 7 или 236 и 8 или 237, соответственно;
(vii) 2-4, 53, 7 или 236 и 8 или 237, соответственно;
(viii) 2-5, 54, 7 или 236 и 8 или 237, соответственно;
(ix) 56-58 и 60-62 соответственно;
(x) 64-66 и 68-70 соответственно;
(xi) 64-66, 73 или 74, 69 и 70 соответственно;
(xii) 64-66, 68, 69 и 77 или 78 соответственно;
(xiii) 80-82 и 84-86 соответственно;
(xiv) 88-90 и 92-94, соответственно;
(xv) 96-98 и 101-102 соответственно;
(xvi) 96, 97, 104 и 100-102, соответственно;
(xvii) 106-108 и 169-171, соответственно;
(xviii) 106, любой из 121-126, 108 и 169-171, соответственно;
(xix) 106, 107, 127 или 128 и 169-171, соответственно;
(xx) 106, 107 или любой из 121-126, 108 и 169-171, соответственно;
(xxi) 156-158 и 160-162, соответственно;
(xxii) 106, 123, 127 и 169-171, соответственно;
(xxiii) 2, 17, 25, 6 или 235 или любой из 51-54, 7 или 236 и 8 или 237, соответственно;
(xxiv) 2, 20, 25, 6 или 235 или любой из 51-54, 7 или 236 и 8 или 237, соответственно;
(xxv) 179-181 и 183-185, соответственно;
(xxvi) 195, 180, 181 и 183-185, соответственно;
(xxvii) 195, 197, 181 и 183-185 соответственно;
(xxviii) 195, 199, 181 и 183-185 соответственно;
(xxiv) 195, 197, 201 и 183-185, соответственно;
(xxx) 195, 197, 203 и 183-185 соответственно;
(xxxi) 195, 199, 201 и 183-185, соответственно;
(xxxii) 195, 199, 203 и 183-185 соответственно;
(xxxiii) 179, 180, 181 и 183-185, соответственно;
(xxxiv) 179, 197, 181 и 183-185, соответственно;
(xxxv) 179, 199, 181 и 183-185, соответственно;
(xxxvi) 179, 197, 201 и 183-185, соответственно;
(xxxvii) 179, 197, 203 и 183-185, соответственно;
(xxxviii) 179, 199, 201 и 183-185, соответственно;
(xxxix) 179, 199, 203 и 183-185, соответственно;
(xxxx) 179, 180, 201 и 183-185, соответственно;
(xxxxi) 179, 180, 203 и 183-185, соответственно; или
(xxxxii) 240-242 и 244-246, соответственно.
Вариант осуществления 4. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 1-3, содержащее:
(i) аминокислотную последовательность CDRH1, представленную в SEQ ID NO:106;
(ii) аминокислотную последовательность CDRH2, представленную в SEQ ID NO:121, SEQ ID NO:107, SEQ ID NO:122, SEQ ID NO:123, SEQ ID NO:124, SEQ ID NO:125, или SEQ ID NO:126;
(iii) аминокислотную последовательность CDRH3, представленную в SEQ ID NO:108, SEQ ID NO:127 или SEQ ID NO:128;
(iv) аминокислотную последовательность CDRL1, представленную в SEQ ID NO:169;
(v) аминокислотную последовательность CDRL2, представленную в SEQ ID NO:170; и
(vi) аминокислотную последовательность CDRL3, представленную в SEQ ID NO:171,
где, необязательно, антитело или антигенсвязывающий фрагмент содержит аминокислотные последовательности CDRH1, CDRH2, CDRH3, CDRL1, CDRL2 и CDRL3, представленные в:
(a) SEQ ID NO:106, 121, 108, 169, 170 и 171, соответственно;
(b) SEQ ID NO:106, 121, 127, 169, 170 и 171, соответственно;
(c) SEQ ID NO:106, 121, 128, 169, 170 и 171, соответственно;
(d) SEQ ID NO:106, 107, 108, 169, 170 и 171, соответственно;
(e) SEQ ID NO:106, 107, 127, 169, 170 и 171, соответственно;
(f) SEQ ID NO:106, 107, 128, 169, 170 и 171, соответственно;
(g) SEQ ID NO:106, 122, 108, 169, 170 и 171, соответственно;
(h) SEQ ID NO:106, 122, 127, 169, 170 и 171, соответственно;
(i) SEQ ID NO:106, 122, 128, 169, 170 и 171, соответственно;
(j) SEQ ID NO:106, 123, 108, 169, 170 и 171, соответственно;
(k) SEQ ID NO:106, 123, 127, 169, 170 и 171, соответственно;
(l) SEQ ID NO:106, 123, 128, 169, 170 и 171, соответственно;
(m) SEQ ID NO:106, 124, 108, 169, 170 и 171, соответственно;
(n) SEQ ID NO:106, 124, 127, 169, 170 и 171, соответственно;
(o) SEQ ID NO:106, 124, 128, 169, 170 и 171, соответственно;
(p) SEQ ID NO:106, 125, 108, 169, 170 и 171, соответственно;
(q) SEQ ID NO:106, 125, 127, 169, 170 и 171, соответственно;
(r) SEQ ID NO:106, 125, 128, 169, 170 и 171, соответственно;
(s) SEQ ID NO:106, 126, 108, 169, 170 и 171, соответственно;
(t) SEQ ID NO:106, 126, 127, 169, 170 и 171, соответственно; или
(u) SEQ ID NO:106, 126, 128, 169, 170 и 171, соответственно.
Вариант осуществления 5. Антитело или его антигенсвязывающий фрагмент, содержащее вариабельный домен тяжелой цепи (VH), содержащий аминокислотную последовательность CDRH1, представленную в SEQ ID NO:106, аминокислотную последовательность CDRH2, представленную в SEQ ID NO:121, и аминокислотную последовательность CDRH3, представленную в SEQ ID NO:108, и вариабельный домен легкой цепи (VL), содержащий аминокислотную последовательность CDRL1, представленную в SEQ ID NO:169, аминокислотную последовательность CDRL2, представленную в SEQ ID NO:170, и аминокислотную последовательность CDRL3, представленную в SEQ ID NO:171,
где антитело или антигенсвязывающий фрагмент способно связываться с поверхностным гликопротеином (S) SARS-CoV-2, экспрессируемым на клеточной поверхности клетки-хозяина, на вирионе или на обоих.
Вариант осуществления 6. Антитело или его антигенсвязывающий фрагмент по варианту осуществления 5, способное нейтрализовать инфекцию SARS-CoV-2 на модели инфекции in vitro и/или на животной модели инфекции in vivo и/или у человека.
Вариант осуществления 7. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 1-6, содержащее:
(i) VH, содержащий или состоящий из аминокислотной последовательности, имеющей по меньшей мере 85% идентичности с аминокислотной последовательностью, представленной в любой из SEQ ID NO:113, 1, 9-15, 23, 24, 27, 28-46, 55, 63, 79, 87, 95, 103, 105, 114-120, 129-146, 155, 172, 176-178, 194, 196, 198, 200, 202, 239 и 267, где изменение по сравнению с эталонной аминокислотной последовательностью VH, при его наличии, необязательно ограничено одной или более каркасными областями, и/или изменение содержит одну или более замен аминокислоты, кодируемой зародышевой линией; и/или
(ii) VL, содержащий или состоящий из аминокислотной последовательности, имеющей по меньшей мере 85% идентичности с аминокислотной последовательностью, представленной в любой из SEQ ID NO:168, 5, 47-50, 59, 67, 71-72, 75, 76, 83, 91, 99, 109, 147-150, 159, 182, 190, 234 и 243, где изменение по сравнению с эталонной аминокислотной последовательностью VH, при его наличии, необязательно ограничено одной или более каркасными областями, и/или изменение содержит одну или более замен аминокислоты, кодируемой зародышевой линией.
Вариант осуществления 8. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществлений 1-7, где VH содержит или состоит из любой аминокислотной последовательности VH, представленной в Таблице 2, и где VL содержит или состоит из любой аминокислотной последовательности VL, представленной в Таблице 2, где, необязательно, VH и VL содержат или состоят из аминокислотных последовательностей, представленных в SEQ ID NO:
(i) 113 и 168, соответственно;
(ii) 1 и 5 или 234, соответственно;
(iii) любой из 9-15 и 5 или 234, соответственно;
(iv) 23 или 24 и 5 или 234, соответственно;
(v) 27 и 5 или 234, соответственно;
(vi) любой из 28-46 и 5 или 234, соответственно;
(vii) 1 и любой из 47-50, соответственно;
(viii) любой из 9-15 и любой из 47-50, соответственно;
(ix) 23 или 24 и любой из 47-50, соответственно;
(x) 27 и любой из 47-50, соответственно;
(xi) любой из 28-46 и любой из 47-50, соответственно;
(xii) 55 и 59, соответственно;
(xiii) 63 и 67, соответственно;
(xiv) 63 и 71 или 72, соответственно;
(xv) 63 и 75 или 76, соответственно;
(xvi) 79 и 83, соответственно;
(xvii) 87 и 91, соответственно;
(xviii) 95 и 99, соответственно;
(xix) 103 и 99, соответственно;
(xx) 105 и 168, соответственно;
(xxi) любой из 114-120 или 267 и 168, соответственно;
(xxii) 129 и 168, соответственно;
(xxiii) любой из 130-146 и 168, соответственно;
(xxiv) 105 и любой из 147-150, соответственно;
(xxv) любой из 113-120 и любой из 147-150, соответственно;
(xxvi) любой из 130-146 и любой из 147-150, соответственно;
(xxvii) 155 и 159, соответственно;
(xxviii) 172 и 168, соответственно;
(xxix) 176 или 177 и 5 или 234 или любой из 47-50, соответственно;
(xxx) 178 и 182 или 190, соответственно;
(xxxi) 194 и 182 соответственно;
(xxxii) 196 и 182, соответственно;
(xxxiii) 198 и 182, соответственно;
(xxxiv) 200 и 182, соответственно;
(xxxv) 202 и 182, соответственно; или
(xxxvi) 239 и 243, соответственно.
Вариант осуществления 9. Антитело или его антигенсвязывающий фрагмент, содержащее вариабельный домен тяжелой цепи (VH) и вариабельный домен легкой цепи (VL), где VH содержит или состоит из аминокислотной последовательности, представленной в SEQ ID NO:113, и VL содержит или состоит из аминокислотной последовательности, представленной в SEQ ID NO:168.
Вариант осуществления 10. Антитело или его антигенсвязывающий фрагмент, содержащее вариабельный домен тяжелой цепи (VH) и вариабельный домен легкой цепи (VL), где VH содержит или состоит из аминокислотной последовательности, представленной в любой из SEQ ID NO:105, 114-120, 129-146, 172 и 267, и VL содержит или состоит из аминокислотной последовательности, представленной в SEQ ID NO:168.
Вариант осуществления 11. Антитело или его антигенсвязывающий фрагмент, содержащее вариабельный домен тяжелой цепи (VH) и вариабельный домен легкой цепи (VL), где VH содержит или состоит из аминокислотной последовательности, представленной в SEQ ID NO:79, и VL содержит или состоит из аминокислотной последовательности, представленной в SEQ ID NO:83.
Вариант осуществления 12. Антитело или его антигенсвязывающий фрагмент, содержащее вариабельный домен тяжелой цепи (VH), содержащий CDRH1, CDRH2 и CDRH3, и вариабельный домен легкой цепи (VL), содержащий CDRL1, CDRL2 и CDRL3, где CDRH1, CDRH2 и CDRH3 содержат или состоят из аминокислотных последовательностей, представленных в SEQ ID NO:80-82, соответственно, и CDRL1, CDRL2 и CDRL3 содержат или состоят из аминокислотных последовательностей, представленных в SEQ ID NO:84-86, соответственно,
где антитело или антигенсвязывающий фрагмент способно связываться с поверхностным гликопротеином (S) SARS-CoV-2, экспрессируемым на клеточной поверхности клетки-хозяина, на вирионе SARS-CoV-2 или на обоих.
Вариант осуществления 13. Выделенное антитело или его антигенсвязывающий фрагмент, содержащее вариабельный домен тяжелой цепи (VH) и вариабельный домен легкой цепи (VL), где VH содержит или состоит из аминокислотной последовательности, представленной в SEQ ID NO:105, и VL содержит или состоит из аминокислотной последовательности, представленной в SEQ ID NO:168.
Вариант осуществления 14. Антитело или его антигенсвязывающий фрагмент, содержащее вариабельный домен тяжелой цепи (VH), содержащий CDRH1, CDRH2 и CDRH3, и вариабельный домен легкой цепи (VL), содержащий CDRL1, CDRL2 и CDRL3, где CDRH1, CDRH2 и CDRH3 содержат или состоят из аминокислотных последовательностей, представленных в SEQ ID NO:106-108, соответственно, и CDRL1, CDRL2 и CDRL3 содержат или состоят из аминокислотных последовательностей, представленных в SEQ ID NO:169-171, соответственно,
где антитело или антигенсвязывающий фрагмент способно связываться с поверхностным гликопротеином (S) SARS-CoV-2, экспрессируемым на клеточной поверхности клетки-хозяина, на вирионе SARS-CoV-2 или на обоих.
Вариант осуществления 15. Антитело или его антигенсвязывающий фрагмент, содержащее вариабельный домен тяжелой цепи (VH) и вариабельный домен легкой цепи (VL), где VH содержит или состоит из аминокислотной последовательности, представленной в SEQ ID NO:178, и VL содержит или состоит из аминокислотной последовательности, представленной в SEQ ID NO:182 или SEQ ID NO:190.
Вариант осуществления 16. Антитело или его антигенсвязывающий фрагмент, содержащее вариабельный домен тяжелой цепи (VH), содержащий CDRH1, CDRH2 и CDRH3, и вариабельный домен легкой цепи (VL), содержащий CDRL1, CDRL2 и CDRL3, где CDRH1, CDRH2 и CDRH3 содержат или состоят из аминокислотных последовательностей, представленных в SEQ ID NO:179-181, соответственно, и CDRL1, CDRL2 и CDRL3 содержат или состоят из аминокислотных последовательностей, представленных в SEQ ID NO:183-185, соответственно,
где антитело или антигенсвязывающий фрагмент способно связываться с поверхностным гликопротеином (S) SARS-CoV-2, экспрессируемым на клеточной поверхности клетки-хозяина, на вирионе SARS-CoV-2 или на обоих.
Вариант осуществления 17. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 1-16, которое:
(i) распознает эпитоп в мотиве связывания рецептора (RBM) ACE2 (SEQ ID NO:167) SARS-CoV-2;
(ii) способно блокировать взаимодействие между SARS-CoV-2 (например, RBM SARS-CoV-2) и ACE2;
(ii) способно связываться с S-белком SARS-CoV-2 с большей авидностью, чем с S-белком коронавируса SARS;
(iii) распознает эпитоп, который сохраняется в ACE2 RBM SARS-CoV-2 и в ACE2 RBM коронавируса SARS;
(vi) обладает перекрестной реактивностью в отношении SARS-CoV-2 и коронавируса SARS;
(vii) распознает эпитоп в поверхностном гликопротеине SARS-CoV-2, который не входит в RBM ACE2,
или
(viii) любая комбинация (i)-(vii).
Вариант осуществления 18. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 1-17, способное ингибировать взаимодействие между SARS-CoV-2 и любым одним или более из DC-SIGN, L-SIGN и SIGLEC-1.
Вариант осуществления 19. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 1-18, способное ингибировать взаимодействие между SARS-CoV-2 и любым одним или более из: DC-SIGN; L-SIGN; SIGLEC-1; CD22; CD33; CLEC4M, SIGLEC-16; SIGLEC-15; SIGLEC-14; SIGLEC-12; SIGLEC-11; SIGLEC-10; SIGLEC-9; SIGLEC-8; SIGLEC-7; SIGLEC-6; SIGLEC-5 или любой их комбинации.
Вариант осуществления 20. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 1-19, которое представляет собой изотип IgG, IgA, IgM, IgE или IgD.
Вариант осуществления 21. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 1-20, которое представляет собой изотип IgG, выбранный из IgG1, IgG2, IgG3 и IgG4.
Вариант осуществления 22. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 1-21, которое является человеческим, гуманизированными или химерными.
Вариант осуществления 23. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 1-22, где антитело или антигенсвязывающий фрагмент включает антитело человека, моноклональное антитело, очищенное антитело, одноцепочечное антитело, Fab, Fab', F(ab')2, Fv, scFv или scFab.
Вариант осуществления 24. Антитело или антигенсвязывающий фрагмент по варианту осуществления 23, где scFab содержит:
(i) аминокислотную последовательность, представленную в любой из SEQ ID NO:218-219 и 226-227;
(ii) VL, содержащий аминокислотную последовательность, представленную в SEQ ID NO:168, и VH, содержащий аминокислотную последовательность, представленную в SEQ ID NO: 105 или SEQ ID NO:113; или
(iii) CDRH1, содержащую аминокислотную последовательность, представленную в SEQ ID NO: 106, CDRH2, содержащую аминокислотную последовательность, представленную в SEQ ID NO:107 или 121, CDRH3, содержащую аминокислотную последовательность, представленную в SEQ ID NO:108, CDRL1, содержащую аминокислотную последовательность, представленную в SEQ ID NO:169, CDRL2, содержащую аминокислотную последовательность, представленную в SEQ ID NO:170, и CDRL3, содержащую аминокислотную последовательность, представленную в SEQ ID NO:171.
Вариант осуществления 25. Антитело или антигенсвязывающий фрагмент по варианту осуществления 23, где scFv содержит:
(i) аминокислотную последовательность, представленную в любой из SEQ ID NO:220-221 или 228-229;
(ii) VL, содержащий аминокислотную последовательность, представленную в SEQ ID NO:168, и VH, содержащий аминокислотную последовательность, представленную в SEQ ID NO: 105 или SEQ ID NO:113; или
(iii) CDRH1, содержащую аминокислотную последовательность, представленную в SEQ ID NO:106, CDRH2, содержащую аминокислотную последовательность, представленную в SEQ ID NO:107 или 121, CDRH3, содержащкю аминокислотную последовательность, представленную в SEQ ID NO:108, CDRL1, содержащую аминокислотную последовательность, представленную в SEQ ID NO:169, CDRL2, содержащую аминокислотную последовательность, представленную в SEQ ID NO:170, и CDRL3, содержащую аминокислотную последовательность, представленную в SEQ ID NO:171.
Вариант осуществления 26. Антитело или антигенсвязывающий фрагмент по варианту осуществления 25, где scFv содержит более одного домена VH и более одного домена VL.
Вариант осуществления 27. Антитело или антигенсвязывающий фрагмент по варианту осуществления 26, где scFv содержит:
(i) аминокислотную последовательность, представленную в любой из SEQ ID NO:222-225 или SEQ ID NO:230-233;
(ii) два домена VL, каждый из которых содержит аминокислотную последовательность, представленную в SEQ ID NO:168, и два домена VH, каждый из которых содержит аминокислотную последовательность, представленную в SEQ ID NO:105 или SEQ ID NO:113; или
(iii) два домена VL, каждый из которых содержит CDRL1, содержащую аминокислотную последовательность, представленную в SEQ ID NO:169, CDRL2, содержащую аминокислотную последовательность, представленную в SEQ ID NO:170, и CDRL3, содержащую аминокислотную последовательность, представленную в SEQ ID NO:171, и два домена VH, каждый из которых содержит CDRH1, содержащую аминокислотную последовательность, представленную в SEQ ID NO:106, CDRH2, содержащую аминокислотную последовательность, представленную в SEQ ID NO:107 или 121, CDRH3, содержащую аминокислотную последовательность, представленную в SEQ ID NO:108.
Вариант осуществления 28. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 1-27, где что антитело или антигенсвязывающий фрагмент представляет собой полиспецифическое антитело или антигенсвязывающий фрагмент.
Вариант осуществления 29. Антитело или антигенсвязывающий фрагмент по варианту осуществления 28, где антитело или антигенсвязывающий фрагмент представляет собой биспецифическое антитело или антигенсвязывающий фрагмент.
Вариант осуществления 30. Антитело или антигенсвязывающий фрагмент по варианту осуществления 28 или 29, содержащее:
(i) первый VH и первый VL; и
(ii) второй VH и второй VL,
где первый VH и второй VH являются различными, и каждый независимо содержит аминокислотную последовательность, имеющую по меньшей мере 85% идентичности с аминокислотной последовательностью, представленной в любой из SEQ ID NO:113, 1, 9-15, 23, 24, 27-46, 55, 63, 79, 87, 95, 103, 105, 114-120, 129-146, 155, 172, 176-178, 194, 196, 198, 200, 202, 239 и 267,
где первый VL и второй VL являются различными, и каждый независимо содержит аминокислотную последовательность, имеющую по меньшей мере 85% идентичности с аминокислотной последовательностью, представленной в любой из SEQ ID NO:168, 5, 47-50, 59, 67, 71, 72, 75, 76, 83, 91, 99, 109, 147-150, 159, 182, 190, 234 и 243;
и где первый VH и первый VL вместе образуют первый антигенсвязывающий сайт, и где второй VH и второй VL вместе образуют второй антигенсвязывающий сайт.
Вариант осуществления 31. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 1-30, где антитело или антигенсвязывающий фрагмент дополнительно содержит полипептид Fc или его фрагмент.
Вариант осуществления 32. Антитело или антигенсвязывающий фрагмент по варианту осуществления 31, где полипептид Fc или его фрагмент содержит:
(i) мутацию, усиливающую связывание с FcRn по сравнению с эталонным полипептидом Fc, который не содержит мутацию; и/или
(ii) мутацию, усиливающую связывание с FcγR по сравнению с эталонным полипептидом Fc, который не содержит мутации.
Вариант осуществления 33. Антитело или антигенсвязывающий фрагмент по варианту осуществления 32, где мутация, усиливающая связывание с FcRn, включает: M428L; N434S; N434H; N434A; N434S; M252Y; S254T; T256E; T250Q; P257I; Q311I; D376V; T307A; E380A или любую их комбинацию.
Вариант осуществления 34. Антитело или антигенсвязывающий фрагмент по варианту осуществления 32 или 33, где мутация, усиливающая связывание с FcRn, включает:
(i) M428L/N434S;
(ii) M252Y/S254T/T256E;
(iii) T250Q/M428L;
(iv) P257I/Q311I;
(v) P257I/N434H;
(vi) D376V/N434H;
(vii) T307A/E380A/N434A или
(viii) любую комбинацию (i)-(vii).
Вариант осуществления 35. Антитело или антигенсвязывающий фрагмент по любому из варианту осуществления 32-34, где мутация, усиливающая связывание с FcRn, включает M428L/N434S.
Вариант осуществления 36. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 32-35, где мутация, усиливающая связывание с FcγR, включает S239D; I332E; A330L; G236A или любую их комбинацию.
Вариант осуществления 37. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 32-36, где мутация, усиливающая связывание с FcγR, включает:
(i) S239D/I332E;
(ii) S239D/A330L/I332E;
(iii) G236A/S239D/I332E или
(iv) G236A/A330L/I332E.
Вариант осуществления 38. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 1-37:
которое содержит мутацию, которая изменяет гликозилирование, где мутация, которая изменяет гликозилирование, включает N297A, N297Q или N297G; и/или
которое является агликозилированным и/или афукозилированным.
Вариант осуществления 39. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 31-38, где полипептид Fc содержит мутацию L234A и мутацию L235A.
Вариант осуществления 40. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 1-39, где антитело или антигенсвязывающий фрагмент связывается с S-белком SARS-CoV-2, как измерено с помощью биослойной интерферометрии.
Вариант осуществления 41. Антитело или антигенсвязывающий фрагмент по варианту осуществления 40, где антитело или антигенсвязывающий фрагмент связывается с S-белком SARS-CoV-2 с KD (константа диссоциации) менее чем около 4,5×10-9 M, например, менее чем 4,5×10-9 M.
Вариант осуществления 42. Антитело или антигенсвязывающий фрагмент по варианту осуществления 40 или 41, где антитело или антигенсвязывающий фрагмент связывается с S-белком SARS-CoV-2 с KD менее чем около 1,0×10-10 M, например, менее чем 1,0×10-10 M.
Вариант осуществления 43. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 40-42, где антитело или антигенсвязывающий фрагмент связывается с S-белком SARS-CoV-2 с KD менее чем около 1,0×10-11 M, например, менее чем 1,0×10-11 M.
Вариант осуществления 44. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 40-43, где антитело или антигенсвязывающий фрагмент связывается с S-белком SARS-CoV-2 с KD менее чем около 1×10-12 M, например, менее чем 1×10-12 M.
Вариант осуществления 45. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 1-44, где антитело или антигенсвязывающий фрагмент способно нейтрализовать инфекцию SARS-CoV-2 и/или нейтрализовать инфекцию клетки-мишени с IC50 от около 16 до около 20 мкг/мл.
Вариант осуществления 46. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 1-45, где антитело или антигенсвязывающий фрагмент способно нейтрализовать инфекцию SARS-CoV-2 и/или нейтрализовать инфекцию клетки-мишени с IC50 от около 0,3 до около 0,4 мкг/мл или от около 3 до около 4 нМ.
Вариант осуществления 47. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 1-46, где антитело или антигенсвязывающий фрагмент способно индуцировать антителозависимую клеточно-опосредованную цитотоксичность (ADCC) и/или антителозависимый клеточный фагоцитоз (ADCP) против клетки-мишени, инфицированной SARS-CoV-2.
Вариант осуществления 48. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 40-47, где Fab антитела или антигенсвязывающего фрагмента способен связываться с S-белком SARS-CoV-2 с KD 2,0×10-9 или менее, 1,9×10-9 или менее или 1,8×10-9 или менее.
Вариант осуществления 49. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 1-48, где антитело или антигенсвязывающий фрагмент способно нейтрализовать инфекцию SARS-CoV-2 и не конкурирует с человеческим ACE2 за связывание с S-белком SARS-CoV-2,
где, необязательно, нейтрализация включает нейтрализацию инфекции на модели инфекции in vitro.
Вариант осуществления 50. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 1-49, где антитело или антигенсвязывающий фрагмент способно нейтрализовать инфекцию SARS-CoV-2 с IC50 3,0 нМ, 3,1 нМ, 3,2 нМ, 3,3 нМ, 3,4 нМ, 3,5 нМ, 3,6 нМ, 3,7 нМ, 3,8 нМ, 3,9 нМ или 4,0 нМ.
Вариант осуществления 51. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 47-50, где индуцирование ADCC включает активацию естественной клетки-киллера, которая содержит вариант V158 FcγRIIIa, естественной клетки-киллера, которая содержит вариант F158 FcγRIIIa, или и то, и другое.
Вариант осуществления 52. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 47-51, где ADCP включает вовлечение FcγRIIa и/или FcγRIIIa, экспрессируемых на поверхности фагоцитарной клетки, такой как моноцит, макрофаг или дендритная клетка.
Вариант осуществления 53. Антитело или его антигенсвязывающий фрагмент, которое конкурирует за связывание с поверхностным гликопротеином SARS-CoV-2 с антителом или антигенсвязывающим фрагментом по любому из вариантов осуществления 1-52, где, необязательно, антитело или антигенсвязывающий фрагмент способно ингибировать взаимодействие между SARS-CoV-2 и любым одним или более из DC-SIGN, L-SIGN и SIGLEC-1.
Вариант осуществления 54. Антитело или его антигенсвязывающий фрагмент, которое конкурирует за связывание с поверхностным гликопротеином SARS-CoV-2 с антителом S309 (VH SEQ ID NO:105; VL SEQ ID NO:168) и/или антителом S303 (VH SEQ ID NO:63; VL SEQ ID NO:67), где, необязательно, антитело или антигенсвязывающий фрагмент способно ингибировать взаимодействие между SARS-CoV-2 и любым одним или более из DC-SIGN, L-SIGN и SIGLEC-1.
Вариант осуществления 55. Антитело или его антигенсвязывающий фрагмент, которое конкурирует за связывание с поверхностным гликопротеином SARS-CoV-2 с антителом S304 (VH SEQ ID NO:79; VL SEQ ID NO:81) и/или антителом S315 (VH SEQ ID NO:178; VL SEQ ID NO:182).
Вариант осуществления 56. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 1-55, способное связываться с поверхностным гликопротеином SARS-CoV-2, когда поверхностный гликопротеин SARS-CoV-2 содержится в префузионном тримере.
Вариант осуществления 57. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 1-56, способное связываться с рецептор-связывающим доменом (RBD) поверхностного гликопротеина SARS-CoV-2, когда RBD гликозилирован и/или дегликозилирован, где связывание определяют с использованием поверхностного плазмонного резонанса (SPR), где необязательно:
(1) SPR проводят с использованием прибора Biacore T200 с использованием кинетического подхода с одним циклом, дополнительно необязательно с 3-минутным периодом введения и 20-минутным периодом диссоциации;
(2) антитело или антигенсвязывающий фрагмент захватывают на поверхности;
(3) RBD присутствует в концентрации, составляющей 0,8 нМ, 3,1 нМ, 12,5 нМ, 50 нМ или 200 нМ;
(4) антитело или антигенсвязывающий фрагмент связывается с гликозилированным RBD с KD, составляющей около 2,0 нМ, около 1,9 нМ, около 1,8 нМ, около 1,7 нМ, около 1,6 нМ, около 1,5 нМ, около 1,4 нМ, около 1,3 нМ, около 1,2 нМ, около 1,1 нМ, около 1,0 нМ, около 0,9 нМ, около 0,8 нМ, около 0,7 нМ, около 0,6 нМ, около 0,5 нМ, около 0,4 нМ или около 0,3 нМ, или с KD, составляющей 0,4±0,05 нМ, или с KD, составляющей 0,45±0,05 нМ, или с KD, составляющей 0,5±0,05 нМ, или с KD, составляющей 0,6±0,05 нМ, или с KD, составляющей 0,7±0,05 нМ, или с KD, составляющей 1,7±0,05 нМ; и/или
(5) антитело или антигенсвязывающий фрагмент связывается с дегликозилированным RBD с KD, составляющей около 37,0 нМ, около 8,0 нМ, около 2,0 нМ, около 1,9 нМ, около 1,8 нМ, около 1,7 нМ, около 1,6 нМ, около 1,5 нМ, около 1,4 нМ, около 1,3 нМ, около 1,2 нМ, около 1,1 нМ, около 1,0 нМ или около 0,9 нМ, или с KD, составляющей 37,0±0,05 нМ, или с KD, составляющей 8,0±0,05 нМ, или с KD, составляющей 1,0±0,05 нМ, или с KD, составляющей 0,9±0,05 нМ, или с KD, составляющей 1,3±0,05 нМ, или с KD, составляющей 1,8±0,05 нМ, или с KD, составляющей 1,7±0,05 нМ.
Вариант осуществления 58. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 1-57, способное нейтрализовать инфекцию, вызванную SARS-CoV-2, в клетке легкого человека, где, необязательно, клетка легкого человека включает клетку Calu-3 (клеточная линия рака легкого человека), где, дополнительно, необязательно, антитело или антигенсвязывающий фрагмент имеет IC50 нейтрализации, составляющую около 97 нг/мл.
Вариант осуществления 59. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 1-58, способное связываться с компонентом комплемента C1q человека, где необязательно связывание с C1q определяют с помощью биослойной интерферометрии (BLI), такой как с использованием прибора Octet.
Вариант осуществления 60. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 1-59, способное ингибировать слияние клеток, опосредованное поверхностным гликопротеином SARS-CoV-2.
Вариант осуществления 61. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 1-60, которое не вызывает опосредованного антителом усиления репликации SARS-CoV-2 в мононуклеарной клетке периферической крови (PBMC), или дендритной клетке, полученных от донора человека.
Вариант осуществления 62. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 1-61, содержащее:
(i) CH1-CH3, содержащую или состоящую из аминокислотной последовательности, представленной в SEQ ID NO:173 или 175; и/или
(ii) CL, содержащую или состоящую из аминокислотной последовательности, представленной в SEQ ID NO:174 или SEQ ID NO:193.
Вариант осуществления 63. Выделенное антитело или его антигенсвязывающий фрагмент, содержащее вариабельный домен тяжелой цепи (VH), содержащий аминокислотную последовательность области, определяющей комплементарность (CDR) H1, представленную в SEQ ID NO:106, аминокислотную последовательность CDRH2, представленную в SEQ ID NO:121, и аминокислотную последовательность CDRH3, представленную в SEQ ID NO:108, и вариабельный домен легкой цепи (VL), содержащий аминокислотную последовательность CDRL1, представленную в SEQ ID NO:169, аминокислотную последовательность CDRL2, представленную в SEQ ID NO:170, и аминокислотную последовательность CDRL3, представленную в SEQ ID NO:171,
где антитело или антигенсвязывающий фрагмент способно связываться с поверхностным гликопротеином (S) SARS-CoV-2, экспрессируемым на клеточной поверхности клетки-хозяина, на вирионе SARS-CoV-2 или на обоих.
Вариант осуществления 64. Выделенное антитело или антигенсвязывающий фрагмент по любому из варианту осуществления 1-63, способное связываться с поверхностным гликопротеином (S):
(i) SARS-CoV-2 Wuhan-Hu-1 (SEQ ID NO:165);
ii) SARS-CoV-2 B.1.1.7;
iii) SARS-CoV-2 B.1.351;
(iv) SARS-CoV-2, содержащего любую одну или более из следующих мутаций по типу замены относительно SEQ ID NO:165: N501Y; S477N; N439K; L452R; E484K; Y453F; A520S; K417N; K417V; S494P; N501T; S477R; V367F; P384L; A522S; A522V; V382L; P330S; T478I; S477I; P479S; или
(v) любой комбинации (i)-(iv).
Вариант осуществления 65. Выделенное антитело или антигенсвязывающий фрагмент по варианту осуществления 63 или 64, способное нейтрализовать инфекцию SARS-CoV-2:
(i) на модели инфекции in vitro;
(ii) на животной модели инфекции in vivo;
(iii) у человека; или
(iv) любой комбинацией (i)-(iii).
Вариант осуществления 66. Выделенное антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 63-65, где:
(i) VH содержит или состоит из аминокислотной последовательности, имеющей по меньшей мере 85% идентичности с аминокислотной последовательностью, представленной в SEQ ID NO:113; и/или
(ii) VL содержит или состоит из аминокислотной последовательности, имеющей по меньшей мере 85% идентичности с аминокислотной последовательностью, представленной в SEQ ID NO:168.
Вариант осуществления 67. Выделенное антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 63-66, где:
(i) VH содержит или состоит из аминокислотной последовательности, имеющей по меньшей мере 90% идентичности с аминокислотной последовательностью, представленной в SEQ ID NO:113; и/или
(ii) VL содержит или состоит из аминокислотной последовательности, имеющей по меньшей мере 90% идентичности с аминокислотной последовательностью, представленной в SEQ ID NO:168.
Вариант осуществления 68. Выделенное антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 63-67, где:
(i) VH содержит или состоит из аминокислотной последовательности, имеющей по меньшей мере 95% идентичности с аминокислотной последовательностью, представленной в SEQ ID NO:113; и/или
(ii) VL содержит или состоит из аминокислотной последовательности, имеющей по меньшей мере 95% идентичности с аминокислотной последовательностью, представленной в SEQ ID NO:168.
Вариант осуществления 69. Выделенное антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 63-68, где:
(i) VH содержит или состоит из аминокислотной последовательности, имеющей по меньшей мере 99% идентичности с аминокислотной последовательностью, представленной в SEQ ID NO:113; и/или
(ii) VL содержит или состоит из аминокислотной последовательности, имеющей по меньшей мере 99% идентичности с аминокислотной последовательностью, представленной в SEQ ID NO:168.
Вариант осуществления 70. Выделенное антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 63-69, способное ингибировать взаимодействие между:
(i) SARS-CoV-2 и человеческой DC-SIGN;
(ii) SARS-CoV-2 и человеческой L-SIGN;
(iii) SARS-CoV-2 и человеческим SIGLEC-1; или
(iv) любой комбинацией (i)-(iii).
Вариант осуществления 71. Выделенное антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 63-70, где антитело или антигенсвязывающий фрагмент включает антитело человека, моноклональное антитело, очищенное антитело, одноцепочечное антитело, Fab, Fab', F(ab')2, Fv, scFv или scFab.
Вариант осуществления 72. Выделенное антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 63-71, где антитело или антигенсвязывающий фрагмент дополнительно содержит полипептид Fc или его фрагмент.
Вариант осуществления 73. Выделенное антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 63-72, которое представляет собой изотип IgG, IgA, IgM, IgE или IgD.
Вариант осуществления 74. Выделенное антитело или антигенсвязывающий фрагмент по варианту осуществления 72 или 73, где полипептид Fc или его фрагмент содержит:
(i) мутацию, которая усиливает связывание с FcRn по сравнению с эталонным полипептидом Fc, который не содержит мутацию; и/или
(ii) мутацию, которая усиливает связывание с FcγR по сравнению с эталонным полипептидом Fc, который не содержит мутацию.
Вариант осуществления 75. Выделенное антитело или антигенсвязывающий фрагмент по варианту осуществления 74, где мутация, усиливающая связывание с FcRn, включает:
(i) M428L/N434S;
(ii) M252Y/S254T/T256E;
(iii) T250Q/M428L;
(iv) P257I/Q311I;
(v) P257I/N434H;
(vi) D376V/N434H;
(vii) T307A/E380A/N434A или
(viii) любую комбинацию (i)-(vii).
Вариант осуществления 76. Выделенное антитело или антигенсвязывающий фрагмент по варианту осуществления 75, где мутация, усиливающая связывание с FcRn, включает M428L/N434S.
Вариант осуществления 77. Выделенное антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 74-76, где мутация, усиливающая связывание с FcγR, включает S239D, I332E, A330L, G236A или любую их комбинацию.
Вариант осуществления 78. Выделенное антитело или антигенсвязывающий фрагмент по варианту осуществления 74, где мутация, усиливающая связывание с FcRn, включает:
(i) S239D/I332E;
(ii) S239D/A330L/I332E;
(iii) G236A/S239D/I332E; или
(iv) G236A/A330L/I332E.
Вариант осуществления 79. Выделенное антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 63-78, дополнительно содержащее CH1-CH3, содержащую или состоящую из аминокислотной последовательности, представленной в SEQ ID NO:265 или 266.
Вариант осуществления 80. Выделенное антитело или его антигенсвязывающий фрагмент, содержащее вариабельный домен тяжелой цепи (VH) и вариабельный домен легкой цепи (VL), где VH содержит или состоит из аминокислотной последовательности, представленной в SEQ ID NO:113, и VL содержит или состоит из аминокислотной последовательности, представленной в SEQ ID NO:168.
Вариант осуществления 81. Выделенное антитело или антигенсвязывающий фрагмент по варианту осуществления 80, способное нейтрализовать инфекцию SARS-CoV-2:
(i) на модели инфекции in vitro;
(ii) на животной модели инфекции in vivo;
(iii) у человека; или
(iv) любой комбинации (i)-(iii).
Вариант осуществления 82. Выделенное антитело или антигенсвязывающий фрагмент по варианту осуществления 80 или 81, способное ингибировать взаимодействие между:
i) SARS-CoV-2 и человеческой DC-SIGN;
ii) SARS-CoV-2 и человеческой L-SIGN;
iii) SARS-CoV-2 и человеческим SIGLEC-1; или
(iv) любой комбинацией (i)-(iii).
Вариант осуществления 83. Выделенное антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 80-82, где антитело или антигенсвязывающий фрагмент дополнительно содержит полипептид Fc или его фрагмент.
Вариант осуществления 84. Выделенное антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 80-83, которое представляет собой изотип IgG, IgA, IgM, IgE или IgD.
Вариант осуществления 85. Выделенное антитело или антигенсвязывающий фрагмент по любому из варианту осуществления 83 или 84, где полипептид Fc или его фрагмент содержит:
(i) мутацию, которая усиливает связывание с FcRn по сравнению с эталонным полипептидом Fc, который не содержит мутацию; и/или
(ii) мутацию, которая усиливает связывание с FcγR по сравнению с эталонным полипептидом Fc, который не содержит мутацию.
Вариант осуществления 86. Выделенное антитело или антигенсвязывающий фрагмент по варианту осуществления 85, где мутация, усиливающая связывание с FcRn, включает:
(i) M428L/N434S;
(ii) M252Y/S254T/T256E;
(iii) T250Q/M428L;
(iv) P257I/Q311I;
(v) P257I/N434H;
(vi) D376V/N434H;
(vii) T307A/E380A/N434A или
(viii) любую комбинацию (i)-(vii).
Вариант осуществления 87. Выделенное антитело или антигенсвязывающий фрагмент по варианту осуществления 86, где мутация, усиливающая связывание с FcRn, включает M428L/N434S.
Вариант осуществления 88. Выделенное антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 85-87, где мутация, усиливающая связывание с FcγR, включает S239D, I332E, A330L, G236A или любую их комбинацию.
Вариант осуществления 89. Выделенное антитело или антигенсвязывающий фрагмент по варианту осуществления 88, где мутация, усиливающая связывание с FcγR, включает:
(i) S239D/I332E;
(ii) S239D/A330L/I332E;
(iii) G236A/S239D/I332E или
(iv) G236A/A330L/I332E.
Вариант осуществления 90. Выделенное антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 80-89, дополнительно содержащее CH1-CH3, содержащую или состоящую из аминокислотной последовательности, представленной в SEQ ID NO:265 или 266.
Вариант осуществления 91. Выделенное антитело, которое содержит:
(i) тяжелую цепь, содержащую (i)(1) VH, содержащий или состоящий из аминокислотной последовательности, представленной в SEQ ID NO:113, и (i)(2) CH1-CH3, содержащую или состоящую из аминокислотной последовательности, представленной в SEQ ID NO:173; и
(ii) легкую цепь, содержащую (ii)(1) VL, содержащий или состоящий из аминокислотной последовательности, представленной в SEQ ID NO: 168, и (ii)(2) CL, содержащую или состоящую из аминокислотной последовательности, представленной в SEQ ID NO:174.
Вариант осуществления 92. Выделенное антитело или его антигенсвязывающий фрагмент, способное связываться с поверхностным гликопротеином (S) SARS-CoV-2 и ингибировать взаимодействие между SARS-CoV-2 и человеческой DC-SIGN, человеческой L-SIGN, человеческим SIGLEC-1 или любой их комбинацией.
Вариант осуществления 93. Выделенное антитело, которое содержит:
(i) тяжелую цепь, содержащую (i)(1) VH, содержащий или состоящий из аминокислотной последовательности, представленной в SEQ ID NO:113, и (i)(2) CH1-CH3, содержащую или состоящую из аминокислотной последовательности, представленной в SEQ ID NO:175; и
(ii) легкую цепь, содержащую (ii)(1) VL, содержащий или состоящий из аминокислотной последовательности, представленной в SEQ ID NO: 168, и (ii)(2) CL, содержащую или состоящую из аминокислотной последовательности, представленной в SEQ ID NO:174.
Вариант осуществления 94. Выделенное антитело или его антигенсвязывающий фрагмент, содержащее вариабельный домен тяжелой цепи (VH), содержащий аминокислотную последовательность области, определяющей комплементарность (CDR) H1, представленную в SEQ ID NO:106, аминокислотную последовательность CDRH2, представленную в SEQ ID NO:121, и аминокислотную последовательность CDRH3, представленную в SEQ ID NO:108, и вариабельный домен легкой цепи (VL), содержащий аминокислотную последовательность CDRL1, представленную в SEQ ID NO:169, аминокислотную последовательность CDRL2, представленную в SEQ ID NO:170, и аминокислотную последовательность CDRL3, представленную в SEQ ID NO:171,
где антитело или антигенсвязывающий фрагмент способно связываться с поверхностным гликопротеином (S) SARS-CoV-2, экспрессируемым на клеточной поверхности клетки-хозяина, на вирионе SARS-CoV-2 или на обоих.
Вариант осуществления 95. Выделенное антитело или антигенсвязывающий фрагмент по варианту осуществления 94, способное нейтрализовать инфекцию SARS-CoV-2:
(i) на модели инфекции in vitro;
(ii) на животной модели инфекции in vivo;
(iii) у человека; или
(iv) любой комбинации (i)-(iii).
Вариант осуществления 96. Антитело или антигенсвязывающий фрагмент по варианту осуществления 94 или 95, где:
(i) VH содержит или состоит из аминокислотной последовательности, имеющей по меньшей мере 85% идентичности с аминокислотной последовательностью, представленной в SEQ ID NO:113; и/или
(ii) VL содержит или состоит из аминокислотной последовательности, имеющей по меньшей мере 85% идентичности с аминокислотной последовательностью, представленной в SEQ ID NO:168.
Вариант осуществления 97. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 94-96, где VH содержит или состоит из аминокислотной последовательности, представленной в SEQ ID NO:113, и VL содержит или состоит из аминокислотной последовательности, представленной в SEQ ID NO:168.
Вариант осуществления 98. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 94-97, которое:
(i) способно связываться с поверхностным гликопротеином SARS-CoV-2 с большей авидностью, чем с S-белком коронавируса SARS;
(ii) обладает перекрестной реактивностью в отношении SARS-CoV-2 и коронавируса SARS;
(iii) распознает эпитоп в поверхностном гликопротеине SARS-CoV-2, который не входит в RBM ACE2; или
(iv) любая комбинация (i)-(iii).
Вариант осуществления 99. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 94-98, которое представляет собой изотип IgG, IgA, IgM, IgE или IgD и предпочтительно представляет собой изотип IgG, выбранный из IgG1, IgG2, IgG3 и IgG4.
Вариант осуществления 100. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 94-99:
(i) которое является человеческим, гуманизированным или химерным;
(ii) где антитело или антигенсвязывающий фрагмент включает антитело человека, моноклональное антитело, очищенное антитело, одноцепочечное антитело, Fab, Fab', F(ab')2, Fv, scFv или scFab; и/или
(iii) где антитело или антигенсвязывающий фрагмент представляет собой полиспецифическое антитело или антигенсвязывающий фрагмент, где, необязательно, антигенсвязывающий фрагмент представляет собой биспецифическое антитело или антигенсвязывающий фрагмент.
Вариант осуществления 101. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 94-100, где антитело или антигенсвязывающий фрагмент дополнительно содержит полипептид Fc или его фрагмент.
Вариант осуществления 102. Антитело или антигенсвязывающий фрагмент по варианту осуществления 101, где полипептид Fc или его фрагмент содержит:
(1) мутацию, усиливающую связывание с FcRn по сравнению с эталонным полипептидом Fc, который не содержит мутацию, где необязательно мутация, усиливающаяет связывание с FcRn, включает M428L, N434S, N434H, N434A, N434S, M252Y, S254T, T256E, T250Q, P257I, Q311I, D376V, T307A, E380A или любую их комбинацию, где дополнительно необязательно мутация, усиливающая связывание с FcRn, включает: (i) M428L/N434S; (ii) M252Y/S254T/T256E; (iii) T250Q/M428L; (iv) P257I/Q311I; (v) P257I/N434H; (vi) D376V/N434H; (vii) T307A/E380A/N434A; или (viii) любую комбинацию (i)-(vii) и/или
(2) мутацию, усиливающую связывание с FcγR по сравнению с эталонным полипептидом Fc, который не содержит мутацию, где необязательно мутация, усиливающая связывание с FcγR, включает S239D, I332E, A330L, G236A или любую их комбинацию, где дополнительно необязательно, мутация, усиливающая связывание с FcγR, включает: (i) S239D/I332E; (ii) S239D/A330L/I332E; (iii) G236A/S239D/I332E; или (iv) G236A/A330L/I332E.
Вариант осуществления 103. Антитело или антигенсвязывающий фрагмент по варианту осуществления 102, где мутация, усиливающая связывание с FcRn, включает M428L/N434S, и/или мутация, усиливающая связывание с FcγR, включает G236A/A330L/I332E.
Вариант осуществления 104. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 94-103, содержащее:
(i) CH1-CH3, имеющую 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентичности с SEQ ID NO:173 или 175; и/или
(ii) CL, содержащую аминокислотную последовательность, имеющую 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентичности с аминокислотной последовательностью SEQ ID NO:174.
Вариант осуществления 105. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 94-104, которое содержит мутацию, изменяющую гликозилирование антитела или антигенсвязывающего фрагмента, где мутация, изменяющая гликозилирование антитела или антигенсвязывающего фрагмента, включает N297A, N297Q или N297G, и/или где антитело или антигенсвязывающий фрагмент является агликозилированным и/или афукозилированным.
Вариант осуществления 106. Антитело или антигенсвязывающий фрагмент по любому из пп. 94-105, где антитело или антигенсвязывающий фрагмент связывается с поверхностным гликопротеином SARS-CoV-2 с KD менее чем около 4,5×10-9 М, менее чем около 5×10-9 М, менее чем около 1×10-10 М, менее чем около 5×10-10 М, менее чем около 1×10-11 М, менее чем около 5×10-11 М или менее чем около 1×10-12 М, как измерено с помощью биослойной интерферометрии, где необязательно антитело или антигенсвязывающий фрагмент связывается с поверхностным гликопротеином SARS-CoV-2 с KD менее чем 1×10-12 М, как измерено с помощью биослойной интерферометрии (например, путем иммобилизации антитела или его антигенсвязывающего фрагмента на сенсорах и погружения сенсоров в лунки, содержащие различные концентрации SARS-CoV-2 или RBD, регистрируя кинетику связывания антитела во время фазы ассоциации, после чего сенсоры погружаются в буфер, не наблюдая отделяя кинетики антитела SARS-CoV-2 или RD во время фазы диссоциации.Биосенсоры белка A (Pall ForteBio) можно использовать для иммобилизации рекомбинантных антител при 2,7 мкг/мл в течение 1 минуты, после этапа гидратации в течение 10 минут с помощью буфера для кинетического анализа (KB; 0,01% BSA (бычий сывороточный альбумин) без эндотоксинов, 0,002^ Tween-20 (твин), 0,005% NaN3 в PBS). Кривые ассоциации могут быть зарегестрированы в течение 5 минут путем инкубации сенсоров, покрытых антителами, с различными концентрациями RBD SARS-CoV-1 (Sino Biological) или RBD SARS-CoV-2 (полученный собственными силами в клетках Expi-CHO; остатки 331-550 спйк-белка из BetaCoV/Wuhan-Hu-1/2019, кат. № MN908947). Исследованная концентрация SARS-CoV-2 или RBD может составлять 10 мкг/мл, затем серийно разбавлятьтся 1:2,5 . Диссоциацию можно зарегистрировать в течение 9 минут, перемещая сенсоры в лунки, содержащие KB. Аффинности, представленные значениями KD, могут быть рассчитаны с использованием глобальной модели соответствия (Octet). Использовалось оборудование Octet Red96 (ForteBio).
Вариант осуществления 107. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 94-106, где антитело или антигенсвязывающий фрагмент способно индуцировать антителозависимую клеточно-опосредованную цитотоксичность (ADCC) и/или антителозависимый клеточный фагоцитоз (ADCP) против клетки-мишени, инфицированной SARS-CoV-2,
где необязательно индуцирующая ADCC включает активацию естественной клетки-киллера, которая содержит вариант FcγRIIIa V158, естественной клетки-киллера, которая содержит вариант FcγRIIIa F158, или и то и другое, и/или индуцирующая ADCP включает вовлечение FcγRIIa, экспрессируемого на поверхности фагоцитарной клетки, такой как моноцит, макрофаг или дендритная клетка.
Вариант осуществления 108. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 94-107, где антитело или антигенсвязывающий фрагмент представляет собой Fab, и Fab способен связываться с поверхностным гликопротеином SARS-CoV-2 с KD 2,0×10-9 M или менее, 1,9×10-9 M или менее или 1,8×10-9 M или менее, как измерено с помощью биослойной интерферометрии.
Вариант осуществления 109. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 94-108, где антитело или антигенсвязывающий фрагмент способно нейтрализовать инфекцию SARS-CoV-2 и не конкурирует с человеческим ACE2 за связывание с S-белком SARS-CoV-2.
Вариант осуществления 110. Антитело или его антигенсвязывающий фрагмент, которое конкурирует за связывание с поверхностным гликопротеином SARS-CoV-2 с антителом или антигенсвязывающим фрагментом по любому из вариантов осуществления 94-109.
Вариант осуществления 111. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 94-110, способное связываться с поверхностным гликопротеином SARS-CoV-2, когда поверхностный гликопротеин SARS-CoV-2 содержится в префузионном тримере.
Вариант осуществления 112. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 94-111, способное связываться с рецептор-связывающим доменом (RBD) поверхностного гликопротеина SARS-CoV-2, когда RBD гликозилирован и/или дегликозилирован, где связывание определяют с использованием поверхностного плазмонного резонанса (SPR), где необязательно:
(1) SPR проводят с использованием прибора Biacore T200 с использованием кинетического подхода с одним циклом, дополнительно необязательно с 3-минутным периодом введения и 20-минутным периодом диссоциации;
(2) антитело или антигенсвязывающий фрагмент захватывают на поверхности;
(3) RBD присутствует в концентрации, составляющей 0,8 нМ, 3,1 нМ, 12,5 нМ, 50 нМ или 200 нМ;
(4) антитело или антигенсвязывающий фрагмент связывается с гликозилированым RBD с KD, составляющей около 0,6 нМ, около 0,5 нМ, около 0,4 нМ или около 0,3 нМ, или с KD, составляющей 0,3±0,05 нМ, или с KD, составляющей 0,4±0,05 нМ, или с KD, составляющей 0,45±0,05 нМ, или с KD, составляющей 0,5±0,05 нМ, или с KD, составляющей 0,6±0,05 нМ; и/или
(5) антитело или антигенсвязывающий фрагмент связывается с дегликозилированным RBD с KD, составляющей около 1,6 нМ, около 1,5 нМ, около 1,4 нМ, около 1,3 нМ, около 1,2 нМ, около 1,1 нМ, около 1,0.
Вариант осуществления 113. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 94-112, способное нейтрализовать инфекцию, вызванную SARS-CoV-2, в клетке легкого человека, где, необязательно, клетка легкого человека включает клетку Calu-3, где, дополнительно, необязательно, антитело или антигенсвязывающий фрагмент имеет IC50 нейтрализации, составляющую около 97 нг/мл.
Вариант осуществления 114. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 94-113, способное связываться с компонентом комплемента C1q человека, где необязательно связывание с C1q определяют с помощью биослойной интерферометрии (BLI), такой как с использованием прибора Octet.
Вариант осуществления 115. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 94-114, способное ингибировать слияние клеток, опосредованное поверхностным гликопротеином SARS-CoV-2.
Вариант осуществления 116. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 94-115, которое не вызывает опосредованного антителом усиления репликации SARS-CoV-2 в мононуклеарной клетке периферической крови (PBMC) или дендритной клетке, полученной от донора человека.
Вариант осуществления 117. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 94-116, способное ингибировать взаимодействие между:
i) SARS-CoV-2 и человеческой DC-SIGN;
ii) SARS-CoV-2 и человеческой L-SIGN;
iii) SARS-CoV-2 и человеческим SIGLEC-1; или
(iv) любой комбинацией (i)-(iii).
Вариант осуществления 118. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 94-117, способное связываться с поверхностным гликопротеином:
(i) SARS-CoV-2 Wuhan-Hu-1 (SEQ ID NO:165);
(ii) SARS-CoV-2 B.1.1.7;
(iii) SARS-CoV-2 B.1.351;
(iv) SARS-CoV-2, содержащего любую одну или более из следующих мутаций по типу замены относительно SEQ ID NO:165: N501Y; S477N; N439K; L452R; E484K; Y453F; A520S; K417N; K417V; S494P; N501T; S477R; V367F; P384L; A522S; A522V; V382L; P330S; T478I; S477I; P479S; или
(v) любой комбинации (i)-(iv).
Вариант осуществления 119. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 94-118, где антитело или антигенсвязывающий фрагмент способно нейтрализовать инфекцию SARS-CoV-2 с IC50 3,0 нМ, 3,1 нМ, 3,2 нМ, 3,3 нМ, 3,4 нМ, 3,5 нМ, 3,6 нМ, 3,7 нМ, 3,8 нМ, 3,9 нМ или 4,0 нМ.
Вариант осуществления 120. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 94-119, содержащее:
(i) тяжелую цепь, содержащую (i)(1) VH, содержащий или состоящий из аминокислотной последовательности, представленной в SEQ ID NO:113, и (i)(2) CH1-CH3, содержащую или состоящую из аминокислотной последовательности, представленной в SEQ ID NO:173; и
(ii) легкую цепь, содержащую (ii)(1) VL, содержащий или состоящий из аминокислотной последовательности, представленной в SEQ ID NO: 168, и (ii)(2) CL, содержащую или состоящую из аминокислотной последовательности, представленной в SEQ ID NO:174.
Вариант осуществления 121. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 94-120, содержащее:
(i) тяжелую цепь, содержащую (i)(1) VH, содержащий или состоящий из аминокислотной последовательности, представленной в SEQ ID NO:113, и (i)(2) CH1-CH3, содержащую или состоящую из аминокислотной последовательности, представленной в SEQ ID NO:175; и
(ii) легкую цепь, содержащую (ii)(1) VL, содержащий или состоящий из аминокислотной последовательности, представленной в SEQ ID NO: 168, и (ii)(2) CL, содержащую или состоящую из аминокислотной последовательности, представленной в SEQ ID NO:174.
Вариант осуществления 122. Выделенное антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 94-121, содержащее CH1-CH3, содержащую или состоящую из аминокислотной последовательности, представленной в SEQ ID NO:265 или 266.
Вариант осуществления 123. Выделенный полинуклеотид, кодирующий антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 1-122 или кодирующий VH, тяжелую цепь, VL и/или легкую цепь антитела или антигенсвязывающего фрагмента.
Вариант осуществления 124. Выделенный полинуклеотид по варианту осуществления 123, где полинуклеотид содержит дезоксирибонуклеиновую кислоту (ДНК) или рибонуклеиновую кислоту (РНК), где РНК необязательно включает матричную РНК (мРНК).
Вариант осуществления 125. Выделенный полинуклеотид по варианту осуществления 123 или 124, который является кодон-оптимизированным для экспрессии в клетке-хозяине.
Вариант осуществления 126. Выделенный полинуклеотид по любому из вариантов осуществления 123-125, содержащий полинуклеотид, имеющий по меньшей мере 50% идентичности с полинуклеотидной последовательностью, представленной в любой одной или более из SEQ ID NO:186-189, 191-192, 238, 247, 248-255 и 257-262.
Вариант осуществления 127. Выделенный полинуклеотид по любому из вариантов осуществления 123-126, содержащий полинуклеотидную последовательность, представленную в любой одной или более из SEQ ID NO:249, 250 и 257-262.
Вариант осуществления 128. Рекомбинантный вектор, содержащий полинуклеотид по любому из вариантов осуществления 123-127.
Вариант осуществления 129. Клетка-хозяин, содержащая полинуклеотид по любому из вариантов осуществления 123-127 и/или вектор по варианту осуществления 128, где полинуклеотид является гетерологичным по отношению к клетке-хозяину.
Вариант осуществления 130. В-клетка человека, содержащая полинуклеотид по любому из вариантов осуществления 123-129, где полинуклеотид является гетерологичным по отношению к В-клетке человека и/или где В-клетка человека является иммортализованной.
Вариант осуществления 131. Композиция, содержащая
(i) антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 1-122;
(ii) полинуклеотид по любому из вариантов осуществления 123-127;
(iii) рекомбинантный вектор по варианту осуществления 128;
(iv) клетку-хозяина по варианту осyществления п. 129; и/или
(v) В-клетку человека по варианту осуществление 130
и фармацевтически приемлемое вспомогательное вещество, носитель или разбавитель.
Вариант осуществления 132. Композиция по варианту осуществления 131, содержащая два или более антитела или антигенсвязывающих фрагментов по любому из вариантов осуществления 1-122.
Вариант осуществления 133. Композиция по варианту осуществления 132, содержащая:
(i) первое антитело или антигенсвязывающий фрагмент, содержащее VH, содержащий или состоящий из аминокислотной последовательности, представленной в SEQ ID NO:79, и VL, содержащий или состоящий из аминокислотной последовательности, представленной в SEQ ID NO:83; и
(ii) второе антитело или антигенсвязывающий фрагмент, содержащее VH, содержащий или состоящий из аминокислотной последовательности, представленной в SEQ ID NO:105, и VL, содержащий или состоящий из аминокислотной последовательности, представленной в SEQ ID NO:168.
Вариант осуществления 134. Композиция по варианту осуществления 132, содержащая:
(i) первое антитело или антигенсвязывающий фрагмент, содержащее вариабельный домен тяжелой цепи (VH), содержащий CDRH1, CDRH2 и CDRH3, и вариабельный домен легкой цепи (VL), содержащий CDRL1, CDRL2 и CDRL3, где CDRH1, CDRH2 и CDRH3 содержат или состоят из аминокислотных последовательностей, представленных в SEQ ID NO:80-82, соответственно, и CDRL1, CDRL2 и CDRL3 содержат или состоят из аминокислотных последовательностей, представленных в SEQ ID NO:84-86, соответственно, и
(ii) второе антитело или антигенсвязывающий фрагмент, содержащее вариабельный домен тяжелой цепи (VH), содержащий CDRH1, CDRH2 и CDRH3, и вариабельный домен легкой цепи (VL), содержащий CDRL1, CDRL2 и CDRL3, где CDRH1, CDRH2 и CDRH3 содержат или состоят из аминокислотных последовательностей, представленных в SEQ ID NO:106-108, соответственно, и CDRL1, CDRL2 и CDRL3 содержат или состоят из аминокислотных последовательностей, представленных в SEQ ID NO:169-171, соответственно.
Вариант осуществления 135. Композиция по варианту осуществления 132, содержащая:
(i) первое антитело или антигенсвязывающий фрагмент, содержащее VH, содержащий или состоящий из аминокислотной последовательности, представленной в SEQ ID NO:178, и VL, содержащий или состоящий из аминокислотной последовательности, представленной в SEQ ID NO:182 или SEQ ID NO:190; и
(ii) второе антитело или антигенсвязывающий фрагмент, содержащее VH, содержащий или состоящий из аминокислотной последовательности, представленной в SEQ ID NO:105, и VL, содержащий или состоящий из аминокислотной последовательности, представленной в SEQ ID NO:168.
Вариант осуществления 136. Композиция по варианту осуществления 135, содержащая:
(i) первое антитело или антигенсвязывающий фрагмент, содержащее вариабельный домен тяжелой цепи (VH), содержащий CDRH1, CDRH2 и CDRH3, и вариабельный домен легкой цепи (VL), содержащий CDRL1, CDRL2 и CDRL3, где CDRH1, CDRH2 и CDRH3 содержат или состоят из аминокислотных последовательностей, представленных в SEQ ID NO:179-181, соответственно, и CDRL1, CDRL2 и CDRL3 содержат или состоят из аминокислотных последовательностей, представленных в SEQ ID NO:183-185, соответственно; и
(ii) второе антитело или антигенсвязывающий фрагмент, содержащее вариабельный домен тяжелой цепи (VH), содержащий CDRH1, CDRH2 и CDRH3, и вариабельный домен легкой цепи (VL), содержащий CDRL1, CDRL2 и CDRL3, где CDRH1, CDRH2 и CDRH3 содержат или состоят из аминокислотных последовательностей, представленных в SEQ ID NO:106-108, соответственно, и CDRL1, CDRL2 и CDRL3 содержат или состоят из аминокислотных последовательностей, представленных в SEQ ID NO:169-171, соответственно.
Вариант осуществления 137. Композиция по варианту осуществления 132, содержащая:
(i) первое антитело или антигенсвязывающий фрагмент, содержащее VH, содержащий или состоящий из аминокислотной последовательности, представленной в SEQ ID NO:178, и VL, содержащий или состоящий из аминокислотной последовательности, представленной в SEQ ID NO:182 или SEQ ID NO:190; и
(ii) второе антитело или антигенсвязывающий фрагмент, содержащие VH, содержащий или состоящий из аминокислотной последовательности, представленной в SEQ ID NO:63, и VL, содержащий или состоящий из аминокислотной последовательности, представленной в SEQ ID NO:67, любой из SEQ ID NO:71-71 или любой из SEQ ID NO:75-76.
Вариант осуществления 138. Композиция по варианту осуществления 132, содержащая:
(i) первое антитело или антигенсвязывающий фрагмент, содержащее вариабельный домен тяжелой цепи (VH), содержащий CDRH1, CDRH2 и CDRH3, и вариабельный домен легкой цепи (VL), содержащий CDRL1, CDRL2 и CDRL3, где CDRH1, CDRH2 и CDRH3 содержат или состоят из аминокислотных последовательностей, представленных в SEQ ID NO:179-181, соответственно, и CDRL1, CDRL2 и CDRL3 содержат или состоят из аминокислотных последовательностей, представленных в SEQ ID NO:183-185, соответственно; и
(ii) второе антитело или антигенсвязывающий фрагмент, содержащее вариабельный домен тяжелой цепи (VH), содержащий CDRH1, CDRH2 и CDRH3, и вариабельный домен легкой цепи (VL), содержащий CDRL1, CDRL2 и CDRL3, где CDRH1, CDRH2 и CDRH3 содержат или состоят из аминокислотных последовательностей, представленных в SEQ ID NO:64-66, соответственно, CDRL1 содержит или состоит из аминокислотных последовательностей, представленных в любой из SEQ ID NO:68, SEQ ID NO:73 или SEQ ID NO:74, CDRL2 содержит или состоит из аминокислотных последовательностей, представленных в SEQ ID NO:69, и CDRL3 содержит или состоит из аминокислотных последовательностей, представленных в SEQ ID NO:70, SEQ ID NO:77 или SEQ ID NO:78.
Вариант осуществления 139. Композиция, содержащая (i) антитело или антигенсвязывающий фрагмент по варианту осуществления 8 или 9 и (ii) антитело или антигенсвязывающий фрагмент по варианту осуществления 10 или 11, где композиция способна нейтрализовать инфекцию SARS-CoV-2 с IC50 от около 0,07 до около 0,08 мкг/мл.
Вариант осуществления 140. Композиция, содержащая полинуклеотид по любому из вариантов осуществления 123-127, инкапсулированный в молекулу-носитель, где молекула-носитель необязательно включает липид, носитель, полученный из липида, такой как липосома, твердая липидная наночастица, маслянистая суспензия, субмикронная липидная эмульсия, липидный микропузырь, обратная липидная мицелла, кохлеарная липосома, липидная микротрубочка, липидный микроцилиндр, липидная наночастица (LNP) или наноразмерная платформа.
Вариант осуществления 141. Способ лечения инфекции SARS-CoV-2 у субъекта, включающий введение указанному субъекту эффективного количества:
(i) антитела или антигенсвязывающего фрагмента по любому из вариантов осуществления 1-122;
(ii) полинуклеотида по любому из вариантов осуществления 123-127;
(iii) рекомбинантного вектора по варианту осуществления 128;
(iv) клетки-хозяина по варианту осyществления п. 129;
(v) В-клетки человека по варианту осуществлениея 130 и/или
(vi) композиции по любому из вариантов осуществления 131-140.
Вариант осуществления 142. Способ ингибирования инфекции SARS-CoV-2 у субъекта, включающий введение указанному субъекту эффективного количества:
(i) антитела или антигенсвязывающего фрагмента по любому из вариантов осуществления 1-122;
(ii) полинуклеотида по любому из вариантов осуществления 123-127;
(iii) рекомбинантного вектора по варианту осуществления 128;
(iv) клетки-хозяина по варианту осyществления п. 129;
(v) В-клетки человека по варианту осуществлениея 130 и/или
(vi) композиции по любому из вариантов осуществления 131-140.
Вариант осуществления 143. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 1-122, полинуклеотид по любому из вариантов осуществления 123-127, рекомбинантный вектор по варианту осуществления 128, клетка-хозяин по варианту осуществления 129, В-клетка человека по вариантов осуществления 130 и/или композиция по любому из вариантов осуществления 131-140 для применения в способе лечения инфекции SARS-CoV-2 у субъекта.
Вариант осуществления 144. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 1-122, полинуклеотид по любому из вариантов осуществления 123-127, рекомбинантный вектор по варианту осуществления 128, клетка-хозяин по варианту осуществления 129, В-клетка человека по варианту осуществления 130 и/или композиция по любому из вариантов осуществления 131-140 для применения в способе ингибирования инфекции SARS-CoV-2 у субъекта.
Вариант осуществления 145. Антитело или антигенсвязывающий фрагмент по любому из вариантов осуществления 1-122, полинуклеотид по любому из вариантов осуществления 123-127, рекомбинантный вектор по варианту осуществления 128, клетка-хозяин по варианту осуществления 129, В-клетка человека по варианту осуществления 130 и/или композиция по любому из вариантов осуществления 131-140 в получении лекарственного средства для лечения инфекции SARS-CoV-2 у субъекта.
Вариант осуществления 146. Способ диагностики инфекции SARS-CoV-2 in vitro, включающий:
(i) приведение в контакт образца, полученного от субъекта, с антителом или антигенсвязывающим фрагментом по любому из вариантов осуществления 1-122 и
(ii) обнаружение комплекса, содержащего антиген и антитело или содержащего антиген и антигенсвязывающий фрагмент.
Вариант осуществления 147. Способ по варианту осуществления 146, где образец содержит кровь, выделенную из субъекта.
Вариант осуществления 148. Комбинация или композиция, содержащая:
(i) антитело или антигенсвязывающий фрагмент, содержащее
(i)(a) аминокислотную последовательность CDRH1 GYPFTSYG, аминокислотную последовательность CDRH2 ISTYNGNT или ISTYQGNT, аминокислотную последовательность CDRH3 ARDYTRGAWFGESLIGGFDN; аминокислотную последовательность CDRL1 или QTVSSTS, аминокислотную последовательность CDRL2 GAS и аминокислотную последовательность CDRL3 QHDTSLT; или
(i)(b) аминокислотную последовательность VH, содержащую или состоящую из QVQLVQSGAEVKKPGASVKVSCKASGYPFTSYGISWVRQAPGQGLEWMGWISTYNGNTNYAQKFQGRVTMTTDTSTTTGYMELRRLRSDDTAVYYCARDYTRGAWFGESLIGGFDNWGQGTLVTVSS
или содержащую или состоящую из QVQLVQSGAEVKKPGASVKVSCKASGYPFTSYGISWVRQAPGQGLEWMGWISTYQGNTNYAQKFQGRVTMTTDTSTTGYMELRRLRSDDTAVYYCARDYTRGAWFGESLIGGFDNWGQGTLVTVSS,
и аминокислотную последовательность VL, содержащую или состоящую из EIVLTQSPGTLSLSPGERATLSCRASQTVSSTSLAWYQQKPGQAPRLLIYGASSRATGIPDRFSGSGSGTDFTLTISRLEPEDFAVYYCQQHDTSLTFGGGTKVEIK;
и
(ii) антитело или антигенсвязывающий фрагмент, содержащее
(ii)(a) аминокислотные последовательности VH и VL, представленные в SEQ ID NO:79 и 83, соответственно;
(ii)(b) аминокислотные последовательности CDRH1, CDRH2, CDRH3, CDRL1, CDRL2 и CDRL3, представленные в SEQ ID NO:80-82 и 84-86, соответственно;
(ii)(c) аминокислотные последовательности VH и VL, представленные в SEQ ID NO:178, или 194, или 196, или 198, или 200, или 202 и 182 или 190, соответственно; или
(ii)(d) аминокислотные последовательности CDRH1, CDRH2, CDRH3, CDRL1, CDRL2 и CDRL3, представленные в SEQ ID NO:179 или 195, 180, или 197, или 199, 181, 201 или 203 и 183-185, соответственно.
Вариант осуществления 149. Способ предотвращения, или лечения, или нейтрализации коронавирусной инфекции у субъекта, включающий введение субъекту комбинации или композиции по варианту осуществления 148, где, необязательно, антитело или антигенсвязывающий фрагмент (i) и антитело или антигенсвязывающий фрагмент (ii) вводят параллельно, одновременно или последовательно.
Вариант осуществления 150. Способ предотвращения, или лечения, или нейтрализации коронавирусной инфекции у субъекта, включающий введение субъекту, который получил первое антитело или антигенсвязывающий фрагмент, содержащее:
(a) аминокислотные последовательности VH и VL, представленные в SEQ ID NO:79 и 83, соответственно; или
(b) аминокислотные последовательности CDRH1, CDRH2, CDRH3, CDRL1, CDRL2 и CDRL3, представленные в SEQ ID NO:80-82 и 84-86, соответственно;
и второго антитела или антигенсвязывающего фрагмента, содержащего:
(a) аминокислотную последовательность VH, представленную в SEQ ID NO:105 или 113 и аминокислотную последовательность VL, представленную в SEQ ID NO: 168; или
(b) аминокислоты CDRH1, CDRH2 и CDRH3, представленные в SEQ ID NO: 106-108, соответственно, или SEQ ID NO:106, 121 и 108, соответственно, и аминокислотные последовательности CDRL1, CDRL2 и CDRL3, представленные в SEQ ID NO:169-171, соответственно.
Вариант осуществления 151. Способ предотвращения, или лечения, или нейтрализации коронавирусной инфекции у субъекта, включающий введение субъекту, который получил первое антитело или антигенсвязывающий фрагмент, содержащее:
(a) аминокислотную последовательность VH, представленную в SEQ ID NO:105 или 113 и аминокислотную последовательность VL, представленную в SEQ ID NO: 168; или
(b) аминокислоты CDRH1, CDRH2 и CDRH3, представленные в SEQ ID NO: 106-108, соответственно, или SEQ ID NO:106, 121 и 108, соответственно, и аминокислотные последовательности CDRL1, CDRL2 и CDRL3, представленные в SEQ ID NO:169-171, соответственно;
и второго антитела или антигенсвязывающего фрагмента, содержащеего:
(a) аминокислотные последовательности VH и VL, представленные в SEQ ID NO:79 и 83, соответственно; или
(b) аминокислотные последовательности CDRH1, CDRH2, CDRH3, CDRL1, CDRL2 и CDRL3, представленные в SEQ ID NO:80-82 и 84-86, соответственно.
Вариант осуществления 152. Способ предотвращения, или лечения, или нейтрализации коронавирусной инфекции у субъекта, включающий введение субъекту, который получил первое антитело или антигенсвязывающий фрагмент, содержащее:
(a) аминокислотные последовательности VH и VL, представленные в SEQ ID NO:178, или 194, или 196, или 198, или 200, или 202 и 182 или 190, соответственно; или
(b) аминокислотные последовательности CDRH1, CDRH2, CDRH3, CDRL1, CDRL2 и CDRL3, представленные в SEQ ID NO:179, или 195, 180, или 197, или 199, 181, 201 или 203 и 183-185, соответственно.
и второго антитела или антигенсвязывающего фрагмента, содержащего:
(a) аминокислотную последовательность VH, представленную в SEQ ID NO:105 или 113 и аминокислотную последовательность VL, представленную в SEQ ID NO: 168; или
(b) аминокислоты CDRH1, CDRH2 и CDRH3, представленные в SEQ ID NO: 106-108, соответственно, или SEQ ID NO:106, 121 и 108, соответственно, и аминокислотные последовательности CDRL1, CDRL2 и CDRL3, представленные в SEQ ID NO:169-171, соответственно.
Вариант осуществления 153. Способ предотвращения, или лечения, или нейтрализации коронавирусной инфекции у субъекта, включающий введение субъекту, который получил первое антитело или антигенсвязывающий фрагмент, содержащее:
(a) аминокислотную последовательность VH, представленную в SEQ ID NO:105 или 113 и аминокислотную последовательность VL, представленную в SEQ ID NO: 168; или
(b) аминокислоты CDRH1, CDRH2 и CDRH3, представленные в SEQ ID NO: 106-108, соответственно, или SEQ ID NO:106, 121 и 108, соответственно, и аминокислотные последовательности CDRL1, CDRL2 и CDRL3, представленные в SEQ ID NO:169-171, соответственно;
и второго антитела или антигенсвязывающего фрагмента, содержащеего:
(a) аминокислотные последовательности VH и VL, представленные в SEQ ID NO:178, или 194, или 196, или 198, или 200, или 202 и 182 или 190, соответственно; или
(b) аминокислотные последовательности CDRH1, CDRH2, CDRH3, CDRL1, CDRL2 и CDRL3, представленные в SEQ ID NO:179 или 195, 180, или 197, или 199, 181, 201 или 203 и 183-185, соответственно.















































Примеры
Пример 1
Моноклональные антитела человека, которые связывают спайк-белок SARS-CoV-2
В-клетки донора с предыдущей инфекцией SARS-CoV отсортировали и иммортализовали EBV (вирус Эпштейна - Барр) и подвергали скринингу в 384-луночных планшетах (способ, описанный в европейском патенте EP1597280B1, который включен в настоящий документ посредством ссылки).
Через две недели после иммортализации супернатанты иммортализованных В-клеток тестировали на связывание антитела со спайк-белком SARS-CoV-2 ("S") с помощью способа проточной цитометрии. Вкратце, клетки ExpiCHO трансфицировали S-белком SARS-CoV-2 (штамм BetaCoV/Wuhan-Hu-1/2019) или пустой плазмидой в качестве отрицательного контроля. Были идентифицированы четырнадцать моноклональных антител, которые связывают SARS-CoV-2 S, и были названы как SARS-CoV-2 S300 через SARS-CoV-2 S312 и SARS-CoV-2 S315 соответственно. Данные связывания для SARS-CoV-2 S300 через SARS-CoV-2 S310 показаны на фиг. 4A и 4B (на этих фигурах антитела обозначены как «S300»-«S310» соответственно). Графики, демонстрирующие положительное связывание, обозначены рамками.
Аминокислотные последовательности 3 областей, определяющих комплементарность тяжелой цепи (CDR) и легкой цепи (L) CDR3 некоторых из этих антител, наряду с процентной идентичностью последовательностей гена вариабельной области зародышевой линии (IMGT; imgt.org), представлены в таблице 3.
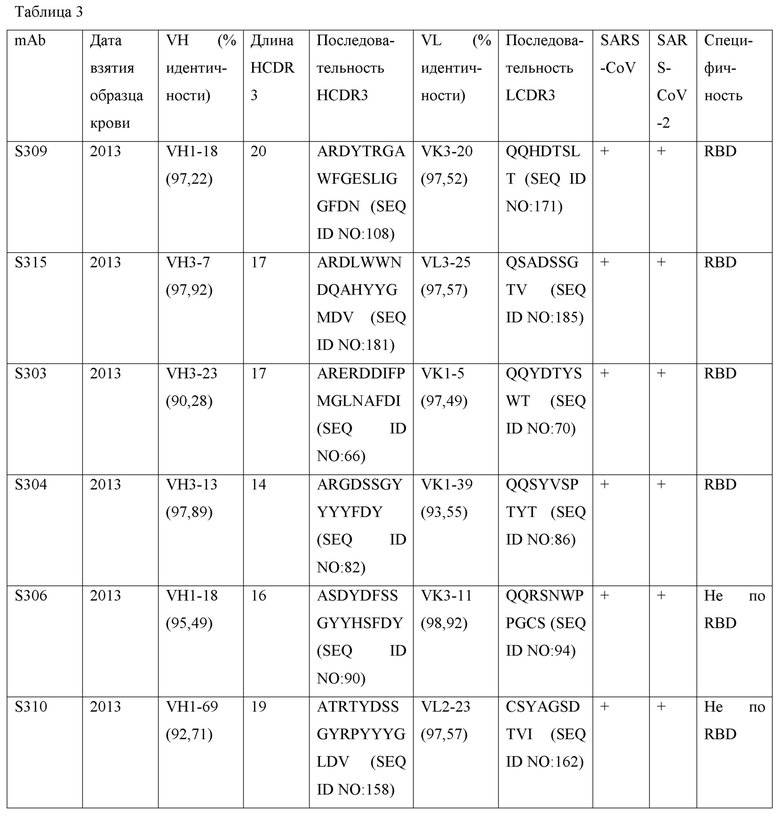
Пример 2
Связывание антител с RBD SARS-CoV-2 с использованием Octet
Биосенсоры стрепавидина (Pall ForteBio) использовали для иммобилизации антитела к Strep Tag II при 3 мкг/мл (клон 5A9F9, биотин, LabForce AG, Muttenz CH) после этапа гидратации в течение 10 минут с кинетическим буфером (KB; 0,01% BSA без эндотоксинов, 0,002^ Tween-20, 0,005% NaN3 в PBS). Затем RBD SARS-CoV-2 с Strep Tag II (полученный собственными силами) загружали в течение 6 мин в концентрации 4 мкг/мл в KB. Антитела из супернатанта В-клеток оставляли ассоциироваться в течение 1620 секунд (27 минут). Для наблюдения за диссоциацией сенсоры перемещали из раствора антитела в KB и контролировали диссоциацию антител.
"S303" mAb содержит аминокислотные последовательности S303-v1 VH и VL, представленные в таблице 2 (SEQ ID NO:63 и 67, соответственно). "S309" mAb содержит аминокислотные последовательности S309-v1 VH и S309-v13 VL, представленные в таблице 2 (SEQ ID NO: 105 и 168, соответственно). Аллели, кодирующие SEQ ID NO:109 и 147-150 из B-клетки S309, определяли как непродуктивные; SEQ ID NO:168 представляет собой продуктивный аллель.
Сравнение кривых связывания для мАт S303 и S309 с RBD SARS-CoV-2 (фиг. 1A и 1B) указывает на то, что S303 имеет как более высокую скорость ассоциации, так и более высокую скорость диссоциации, чем S309, что позволяет предположить, что S309 может связываться с RBD SARS-CoV-2 с более высокой аффинностью.
Пример 3
Оценка связывания антител с RBD SARS-CoV-2 и SARS-CoV-1 с использованием Octet
Если контекст явно не указывает на иное (например, что антитела присутствовали в супернатанте В-клеток или использовали фрагмент Fab антитела), антитела по настоящему изобретению описаны в этом и последующих примерах как рекомбинантно экспрессированные IgG1 человека, в некоторых случаях с аминокислотными мутациями в Fc, как описано в настоящем документе.
Аффинность связывания трех перекрестно-реактивных рекомбинантных антител к SARS-CoV/SARS-CoV-2 (S303 rIgG1, S304 rIgG1, S309 rIgG1) и двух специфических антител к SARS-CoV-1 (S109 rIgG1, S230 rIgG1) исследовали с помощью биослойной интерферометрии (BLI) с использованием Octet. Аффинность измеряли путем иммобилизации антитела на сенсорах и сенсорах погружения в лунки, содержащие различные концентрации RBD.
Кинетику связывания антитела с RBD регистрировали во время фазы ассоциации, после чего датчики погружали в буфер без антитела для наблюдения кинетики отделения антитела от RBD во время фазы диссоциации. Кратко, биосенсоры белка A (Pall ForteBio) использовали для иммобилизации рекомбинантных антител при 2,7 мкг/мл в течение 1 минуты, после этапа гидратации в течение 10 минут с помощью буфера для кинетического анализа (KB; 0,01% BSA без эндотоксинов, 0,002^ Tween-20, 0,005% NaN3 в PBS). Кривые ассоциации регестрировали в течение 5 минут путем инкубации сенсоров, покрытых антителами, с различными концентрациями RBD SARS-CoV-1 (Sino Biological) или RBD SARS-CoV-2 (полученный собственными силами в клетках Expi-CHO; остатки 331-550 спайк-белка из BetaCoV/Wuhan-Hu-1/2019, кат. № MN908947). Самая высокая протестированная концентрация RBD составляла 10 мкг/мл, затем серийно разбавляли 1:2,5 с. Диссоциацию регистрировали в течение 9 минут, перемещая сенсоры в лунки, содержащие KB. Аффинности, представленные значениями KD, рассчитывали с использованием глобальной модели соответствия (Octet). Использовали оборудование Octet Red96 (ForteBio).
На фиг. 6A-6E показаны кривые ассоциации и диссоциации для антител с использованием наивысшей испытанной концентрации RBD (10 мкг/мл). Переключение с раствора RBD на буфер индицируется вертикальной пунктирной линией. Тестировали три перекрестно-реактивных антитела (S303 rIgG1, S304 rIgG1 (VH с SEQ ID NO:79, VL с SEQ ID NO:73), S309 rIgG1 (VH с SEQ ID NO:105, VL с SEQ ID NO:168) и два специфических антитела к SARS-CoV-1 (S230 и S109). Все антитела продемонстрировали сильное связывание с RBD SARS-CoV-1. S230 и S109 не связывались с RBD SARS-CoV-2. Связывание S303 rIgG1, S304 rIgG1 и S309 rIGg1 с RBD SARS-CoV-2 находилось в диапазоне от наномолярного до субпикомолярного, причем S309 rIgG1 демонстрировал наивысшую аффинность. Значения KD указаны под графиками на фиг. 6A-6E. Значения KD представляют собой оценки (KD составляет менее 1,0×10-12M), если связывание антител очень сильное, а диссоциация медленная. Точное значение KD для S309 rIgG1 не может быть измерено с помощью этого анализа, поскольку диссоциация была слишком медленной.
Пример 4
Нейтрализация инфекции SARS-CoV-2
Некомпетентные к репликации вирусы, псевдотипированные геном SARS-CoV-2 (изолят BetaCoV/Wuhan-Hu-1/2019; кат. № MN908947), была получена с использованием способов, описанных ранее (Temperton NJ, и др. (2005) Longitudinally profiling neutralizing antibody response to SARS coronavirus with pseudotypes. Emerg Infect Dis 11(3):411-416). Вкратце, клетки HEK293T/17 котрансфицировали S-экспрессирующей плазмидой S-клеточного SARS-CoV-2 (phCMV1, Genlantis) и вектором комплементарного репортерного гена вирусного генома pNL4-3. Luc+E-R+. Для измерения нейтрализации вирионов, кодирующих люциферазу, псевдотипированных S-белком SARS-CoV-2, как описано ранее, использовали одноцикловый анализ инфекционности (Temperton NJ, и др. (2007) A sensitive retroviral pseudotype assay for influenza H5N1-neutralizing antibodies. Influenza Other Respi Viruses 1(3):105-112.). Вкратце, соответствующие разведения супернатантов культуры, содержащих вирион, предварительно инкубировали при 37 °С в течение 1 часа с антителами в различных концентрациях, а затем смеси вируса и mAb добавляли к клеткам Vero E6, которые были посеяны за день до инфекции. Затем клетки лизировали реагентом Steady-Glo (Promega, E2520), и относительные единицы люминесценции (RLU) в клеточных лизатах определяли на микропланшетном ридере с люминометром (Synergy H1 Hybrid Multi-Mode Reader; Biotek). Снижение инфективности определяли путем сравнения RLU в присутствии и отсутствии антитела и выражали в процентах нейтрализации.
Антитела S300-v1 (VH: SEQ ID NO:1; VL: SEQ ID NO:5), S301, S302, S303-v1 (VH SEQ ID NO:63; VL SEQ ID NO:67), S304 (VH SEQ ID NO:79; VL SEQ ID NO:83), S306 (VH SEQ ID NO:87; VL SEQ ID NO:91), S307 (VH SEQ ID NO:239; VL SEQ ID NO:243), S308-v1, S309 (содержащая последовательность VH S309-v1, представленную в SEQ ID NO: 105, и последовательность VL S309-v13, представленную в SEQ ID NO: 168) и S310 тестировали на эффект нейтрализации (таблица 4, фиг. 2A). Антитела SARS-CoV-2 S300-v1 и SARS-CoV-2 S309 нейтрализовали инфекцию SARS-CoV-2 (фиг. 2A и 2B).
Дополнительные анализы нейтрализации проводили с использованием плазмы от выживших пациентов после SARS CoV-1 и антител SARS-CoV-2 S309, S311, S312, S303-v1 (rIgG1), S304 (rIgG1), S306 (rIgG1), S310 (rIgG1) и S315 (фиг. 3A-3I). На фиг. 3А показана нейтрализующая активность донорской плазмы SARS-CoV. На фиг. 3B-3D и 3I показана нейтрализующая активность супернатанта из В-клеток, продуцирующих S309, S311, S312 и S315, соответственно. На фиг. 3E-3H показана нейтрализующая активность рекомбинантного антитела в различных концентрациях. Используя этот анализ, супернатант, содержащий антитело S309, S311, S312 или S315, нейтрализует инфекцию SARS-CoV-2.
Дополнительные анализы нейтрализации проводили с использованием антител S303, S304, S306, S309 (VH SEQ ID NO:105; VL SEQ ID NO:168), S310 и S315. На фиг. 13 показана нейтрализующая активность этих антител в различных концентрациях против MLV, псевдотипированного SARS-CoV-2. В качестве клеток-мишеней использовали DBT-клетки, стабильно трансфицированные ACE2 (DBT-ACE2). На фиг. 34 показана нейтрализующая активность против MLV, псевдотипированного SARS-CoV-1 этими антителами в различных концентрациях. Дополнительные данные нейтрализации для S304, S309, S304 + S309, S315 и S315 + S309 показаны на фиг. 36 и 37.
Пример 5
Нейтрализация инфекции SARS-CoV-2
Оценивали нейтрализующую активность двух кросс-нейтрализующих антител SARS-CoV-1 и SARS-CoV-2, S304 rIgG1 и S309 (VH: SEQ ID NO:105; VL: SEQ ID NO:168) rIgG1, против вирусов, псевдотипированных SARS-CoV-2 (SARS-CoV-2pp).
Использовали вирус мышиного лейкоза (MLV), псевдотипированный спайк-белком SARS-CoV-2 (SARS-CoV-2pp). В качестве клеток-мишеней использовали DBT-клетки, стабильно трансфицированные ACE2 (DBT-ACE2). SARS-CoV-2pp активировали трипсином TPCK (тозилфенилаланилхлорметилкетон) в концентрации 10 мкг/мл. Активированный SARS-CoV-2pp добавляли к серии разведений антител (начиная с конечной концентрации 50 мкг/мл на антитело, 3-кратное разведение). Антитела тестировали в концентрациях от 50 мкг/мл до 0,02 мкг/мл. Для комбинации S304 rIgG1 и S309 rIgG1 начальные концентрации составляли 50 мкг/мл для каждого антитела, то есть общая начальная концентрация антитела составляла 100 мкг/мл. Клетки DBT-ACE2 добавляли к смесям антитело-вирус и инкубировали в течение 48 часов. Люминесценцию измеряли после аспирации супернатанта клеточной культуры и добавления устойчивого субстрата GLO (Promega).
В этом анализе S309 rIgG1 демонстрировал нейтрализацию IC50 инфекции 0,37 мкг/мл, а S304 rIgG1 демонстрировал IC50 приблизительно 17 мкг/мл. Комбинация этих двух антител продемонстрировала IC50 0,077 мкг/мл. См. фиг. 7 и таблицу 5.
Дальнейшие анализы нейтрализации проводили с использованием той же процедуры для рекомбинантных моноклональных антител S309 и S315, по отдельности и в комбинации. В этом анализе S309 демонстрировал IC50 1,091 мкг/мл, а S315 демонстрировал IC50 25,1 мкг/мл. Комбинация обоих этих антител демонстрировала IC50 0,3047 мкг/мл. См. фиг. 23 и таблицу 6.
Пример 6
Реактивность человеческих моноклональных антител против SARS-CoV и SARS-CoV-2
Реактивность дополнительных человеческих mAb "S311" и "S312" против субъедицы S1 спайк-белка и RBD белка SARS-CoV и SARS-CoV-2 оценивали с помощью твердофазного иммуноферментного анализа (ELISA).
96-луночные планшеты покрывали субъединицей S1 рекомбинантного спайк-белка SARS-CoV-2 (Sino Biological), RBD SARS-CoV-2 (Sino Biological или полученный собственными силами; остатки 331-550 спайка-белка из BetaCoV/Wuhan-Hu-1/2019, кат. № MN908947), субъединицей S1 рекомбинантного спайк-белка SARS-CoV Spike S1 (Sino Biological) или SARS-CoV RBD (Sino Biological).
Лунки промывали и блокировали PBS+1%BSA в течение 1 часа при комнатной температуре, а затем инкубировали с серийно разведенными mAb в течение 1 часа при комнатной температуре. Связанные mAb обнаруживали путем инкубации конъюгированных с щелочной фосфатазой козьих антител против IgG человека (Southern Biotechnology: 2040-04) в течение 1 часа при комнатной температуре и проявляли с помощью 1 мг/мл п-нитрофенилфосфатного субстрата в 0,1 М глициновом буфере (pH 10,4) в течение 30 минут при комнатной температуре. Значения оптической плотности (OD) измеряли при длине волны 405 нм в ридере ELISA (спектрофотометр Powerwave 340/96, BioTek).
Результаты показаны на фиг. 5A (SARS-CoV-2 S311) и фиг. 5B (SARS-CoV-2 S312).
Дальнейшие анализы проводили для исследования реактивности вариантов антител, сконструированных из S300, S305 или S307 к RBD SARS-CoV-2 и SARS-CoV-1, используя ту же процедуру, описанную выше в этом примере. Результаты показаны на фиг. 38A-38D. Антитело "S300 V4-rIgG1", как показано на фиг. 38C, содержит VH, содержащий аминокислотную последовательность SEQ ID NO:1, и VL (Vκ), содержащий аминокислотную последовательность SEQ ID NO:234. Антитело "S307 V3-rIgG1", как показано на фиг. 38D, содержит VH, содержащий аминокислотную последовательность SEQ ID NO: 239, и VL (Vκ), содержащий аминокислотную последовательность SEQ ID NO:243.
Пример 7
Нейтрализация инфекции SARS-CoV-2 с помощью S309 и S315
Определяли нейтрализующую активность рекомбинантных антител S309 (VH: SEQ ID NO:105; VL SEQ ID NO:168) rIgG1-MLNS и S315 rIgG1-MLNS против вирусов, псевдотипированных SARS-CoV-2 (SARS-CoV-2pp). Эти рекомбинантные антитела содержали мутации M428L и N434S в домене Fc (см., например, Zalevsky и др., Nat. Biotechnol. 28(2):157-159 (2010); эта комбинация мутаций Fc также упоминается как "MLNS" или "LS" в настоящем описании, в том числе на фигурах).
Использовали вирус мышиного лейкоза (MLV), псевдотипированный спайк-белком SARS-CoV-2 (SARS-CoV-2pp). В качестве клеток-мишеней использовали DBT-клетки, стабильно трансфицированные ACE2 (DBT-ACE2). SARS-CoV-2pp активировали трипсином TPCK в концентрации 10 мкг/мл. Активированный SARS-CoV-2pp добавляли к серии разведений тестируемого антитела. Клетки DBT-ACE2 добавляли к смесям антитело-вирус и инкубировали в течение 48 часов. Люминесценцию измеряли после аспирации супернатанта клеточной культуры и добавления устойчивого субстрата GLO (Promega). Сигнал люциферазы инфицированных клеток использовали для расчета процента нейтрализации по сравнению с контролем без антител.
S309 rIgG1 MLNS ("S309-rIgG1-LS" на фиг. 9) демонстрировало IC50 нейтрализации инфекции, составляющей приблизительно 3,9 нМ, а S315 rIgG1 MLNS ("S315-rIgG1-LSv1" на фиг. 9) демонстрировало IC50, составляющей приблизительно 111,7 мМ. См. фиг. 9.
Нейтрализующую активность S309-rFab сравнивали с активностью полноразмерного S309 rIgG1 MLNS ("S309-rIgG1-LS" на фиг. 10). Полноразмерное S309 rIgG-LS демонстрировало IC50 3,821 нМ, в то время как S309-rFab демонстрировало IC50 3,532 нМ. См. фиг. 10.
Пример 8
Реактивность антител против RBD SARS-CoV-1, RBD SARS-CoV-2 и эктодоменов различных коронавирусов
Реактивность моноклональных антител против RBD SARS-CoV-1 и SARS-CoV-2 и спайк-белков SARS-CoV-1, SARS-CoV-2, OC43 и MERS коронавируса изучали с помощью твердофазного иммуноферментного анализа (ELISA). 384-луночные неглубокие планшеты для твердофазного ELISA покрывали стабилизированным префузионным тримером спайк-белка SARS-CoV-1, SARS-CoV-2, OC43 или MERS в концентрации 1 мкг/мл, или RBD SARS-CoV-2 (полученный собстивенными силами; остатки 331-550 спайк-белка BetaCoV/Wuhan-Hu-1/2019, кат. № MN908947) в концентрации 10 мкг/мл, или RBD SARS-CoV-1 (Sino Biological) в концентрации 1 мкг/мл.
Лунки промывали и блокировали PBS+1% BSA в течение 1 часа при комнатной температуре, а затем инкубировали с серийно разведенными антителами в течение 1-2 часов при комнатной температуре. Антитела тестировали в диапазоне концентраций от 5 до 0,00028 мкг/мл. Планшеты промывали и выявляли связанные антитела путем инкубации конъюгированного с щелочной фосфатазой козьего антитела против IgG человека (Southern Biotechnology: 2040-04) в течение 1 часа при комнатной температуре с последующим развитием цвета с использованием 1 мг/мл п-нитрофенилфосфатного субстрата (Sigma-Aldrich 71768) в 0,1 М глициновом буфере (pH 10,4) в течение 30 минут при комнатной температуре. Оптическую плотность (OD) измеряли при длине волны 405 нм в ридере ELISA (спектрофотометр Powerwave 340/96, BioTek).
Результаты анализа ELISA показаны на фиг. 8A-8K и 18A-18J. Рекомбинантные антитела, некоторые из которых несут мутации Fc MLNS, показаны с rIgG1.
Пример 9
Связывание антител с о спайк-белком SARS-CoV-1 и SARS-CoV-2
Клетки ExpiCHO трансфицировали phCMV1-SARS-CoV-2-S, SARS-spike_pcDNA.3 (штамм SARS) или пустой phCMV1 с использованием Expifectamine CHO Enhancer. Через два дня после трансфекции клетки собирали для иммуноокрашивания антителами. Для обнаружения использовали меченное Alexa647 вторичное антитело против Fc IgG человека. Связывание моноклонального антитела с трансфицированными клетками анализировали с помощью проточной цитометрии с использованием клеточного анализатора ZE5 (Biorard) и программного обеспечения FlowJo (TreeStar). Положительное связывание определяли дифференциальным окрашиванием трансфектантов CoV-S по сравнению с имитацией трансфектантов. Антитело S309 (VH SEQ ID NO:105; VL SEQ ID NO:168) исследовали с помощью проточной цитометрии в концентрации 10 мкг/мл на способность окрашивать клетки ExpiCHO, экспрессирующие S-белок SARS-CoV-1 или SARS-CoV-2. Сложенные гистограммы графиков проточной цитометрии демонстрируют дозозависимое связывание антитела S309 с S-белком SARS-CoV или SARS-CoV-2. Результаты приведены на фиг. 11.
Связывание моноклональных антител S303, S304, S306, S309, S310, S315, S110, S124, S230 и S109 (все экспрессировались как rIgG1) с S-белком SARS-CoV-1 и S-белком SARS-CoV-2 измеряли с помощью проточной цитометрии. Результаты показаны на фиг. 12A, 12B, 40A и 40B. Восемь из тестируемых антител продемонстрировали значения EC50 в диапазоне от 1,4 нг/мл до 6100 нг/мл для связывания S-белка SARS-CoV-2 и от 0,8 нг/мл до 254 нг/мл для связывания S-белка SARS-CoV-1.
Дополнительные анализы связывания с использованием той же процедуры проводили для S309 и четырех сконструированных вариантов S309, несущих различные мутации в VH (N55Q, W50F, W105F или W50F + G56A + W105F). Результаты приведены на фиг. 27. Значения EC50 для каждого антитела, протестированного в этих анализах, показаны в таблице 7; номера, заключенные в скобки в столбце «Антитело» в таблице 7, соответствуют обозначениям на фиг. 27.
Дополнительные анализы связывания с использованием той же процедуры проводили с использованием антител S303, S304, S306, S309, S310, S315 и сравнительных антител S109, S110, S124 и S230. Результаты показаны на фиг. 33A и 42A (связывание с S-белком SARS-CoV-2) и 33B и 42B (связывание с S-белком SARS-CoV-1). MFI: средняя интенсивность флуоресценции, измеренная с помощью проточной цитометрии.
Такой же анализ проводили с использованием рекомбинантных антител S300 и S307. Результаты для антитела S300-rIgG1, которое содержит VH, содержащий аминокислотную последовательность SEQ ID NO:1, и VL (Vκ), содержащий аминокислотную последовательность SEQ ID NO:234, показаны на фиг. 39A. Результаты для антитела S307-rIgG1, которое содержит VH, содержащий аминокислотную последовательность SEQ ID NO: 239, и VL (Vκ), содержащий аминокислотную последовательность SEQ ID NO:24, показаны на фиг. 39B.
Пример 10
Связывание антител S309, S303, S304 и S315 с RBD SARS-CoV-2 и SARS-CoV-1
Аффинность рекомбинантных антител S309, S303, S304 и S315 к RBD CoV-1 и CoV-2 проверяли с помощью биослойной интерферометрии (BLI; Octet). Вкратце, His-меченную RBD SARS-CoV-1 или SARS-CoV-2 загружали в концентрации 3 мкг/мл в кинетическом буфере (KB) в течение 15 минут на биосенсоры против HIS (HIS2) (Molecular Devices, ForteBio). Ассоциацию полноразмерных антител проводили в KB при 15 мкг/мл в течение 5 минут. Ассоциацию Fab-фрагментов проводили в KB при 5 мкг/мл в течение 5 минут. Диссоциацию в KB измеряли в течение 10 минут. Аффинности, представленные значениями KD, рассчитывали с использованием глобальной модели соответствия (Octet). Использовали оборудование Octet Red96 (ForteBio).
На фиг. 14A-14D показаны кривые ассоциации и диссоциации для S309, S303, S304 и S315, соответственно. Каждое из этих антител связывается с RBD SARS-CoV-2 и SARS-CoV-1 с аффинностью от наномолярной до субпикомолярной. На фиг. 20A и 20B показаны кривые ассоциации и диссоциации для IgG S309 и Fab S309, соответственно. На этих фигурах вертикальной пунктирной линией обозначен переход от раствора антитела (или Fab) к буферу.
Пример 11
Связывание S309 IgG и S309 Fab с тримером эктодомена S-белка SARS-CoV-2 и RBD
Определение аффинности и авидности IgG1 и Fab-фрагмента: биотинилированный RBD SARS-CoV-2 (полученный собственными силами; аминокислотные остатки 331-550 спайк-белка из BetaCoV/Wuhan-Hu-1/2019, кат. № MN908947, биотинилированный EZ-Link NHS-PEG4-Biotin от ThermoFisher) и биотинилированный SARS-CoV-2 2P S с avi-меткой загружали при 7,5 мкг/мл в кинетическом буфере (KB; 0,01% BSA без эндотоксинов, 0,002% Tween-20, 0,005% NaN3 в PBS) в течение 8 минут на биосенсоры стрептавидина (Molecular Devices, ForteBio). Ассоциацию IgG1 и Fab с мишенью проводили в KB при 100, 33, 11, 3,6, 1,2 нМ в течение 5 минут. Диссоциацию в KB измеряли в течение 10 минут. Значения KD рассчитывали с использованием глобальной модели соответствия 1:1 (Octet).
Результаты показаны на фиг. 41A и 41B. В этом анализе IgG S309 связывался с RBD SARS-CoV-2 и с тримером S-эктодомена с субпикомолярной и пикомолярной авидностью, соответственно. S309 Fab, связанный как с RBD SARS-CoV-2, так и с тримером S-эктодомена с наномолярной и субнаномолярной аффинностью.
Пример 12
Конкурентное связывание антител с RBD SARS-CoV-1 или SARS-CoV-2
Конкурентное связывание пар моноклональных антител с RBD SARS-CoV-1 или RBD SARS-CoV-2 измеряли для идентификации соответствующих сайтов связывания антител.
Биосенсоры стрепавидина (Pall ForteBio) использовали для иммобилизации антитела к Strep Tag II при 3 мкг/мл (клон 5A9F9, биотин, LabForce AG, Muttenz CH) после этапа гидратации в течение 10 минут с кинетическим буфером (KB; 0,01% BSA без эндотоксинов, 0,002^ Tween-20, 0,005% NaN3 в PBS). Затем RBD SARS-CoV-1 или SARS-CoV-2 со Strep Tag II (полученный собственными силами) загружали на 6 минут в концентрации 4 мкг/мл в KB. Первому антителу давали ассоциироваться в течение определенного периода времени, а затем второму антителу давали ассоциироваться в течение 7 минут (420 секунд). На фиг. 15A показана конкуренция пар антител за связывание с RBD SARS-CoV-1. На фиг. 15B показана конкуренция пар антител за связывание с RBD SARS-CoV-2. Пунктирные вертикальные линии на фиг. 15A и 15B указывают на переход от первого антитела, указанного слева от матрицы, ко второму антителу, указанному в верхней части матрицы. Используя эти и другие данные, были идентифицированы четыре антигенные области или сайта (I-IV на фиг. 15A и 15B).
Пример 13
Интерференция с RBD: связывание ACE2 человека
Измеряли способность антител препятствовать связыванию RBD с человеческим ACE2. ACE2-His (Bio-Techne AG) загружали в течение 30 минут при 5 мкг/мл в кинетическом буфере (KB) на биосенсоры анти-HIS (HIS2) (молекулярные устройства-ForteBio) Fc кроличьего RBD SARS-CoV-1 или Fc мышиного RBD SARS-CoV-2 (Sino Biological Europe GmbH) при 1 мкг/мл ассоциировали в течение 15 минут после предварительной инкубации с антителом или без антитела при 30 мкг/мл в течение 30 минут. Диссоциацию контролировали в течение 5 минут. На фиг. 16 показаны данные, полученные с использованием антитела S309 или S230. На фиг. 19А и 19В показаны данные, полученные с использованием антител S304, S303 или S230 (фиг. 19А) или RBD и антитела S315 (фиг. 19В). Вертикальная пунктирная линия на каждой из фиг. 16, 19A и 19B указывает на начало загрузки RBD с антителом или без антитела.
Пример 14
Эффекторная функция антител
Опосредованная естественными киллерами (NK) антителозависимая клеточная цитотоксичность (ADCC) может способствовать контролю вируса путем уничтожения инфицированных клеток, презентирующих вирусный белок на своей поверхности. Чтобы исследовать способность антител использовать эту функцию, ADCC исследовали in vitro с использованием человеческих NK-клеток (выделенных из свежей крови здоровых доноров с использованием набора MACSxpress NK Isolation Kit (Miltenyi Biotec, кат. № 130-098-185)) в качестве эффекторных клеток и трансфицированных S-белком SARS-CoV-2 клеток ExpiCHO в качестве клеток-мишеней. Клетки-мишени инкубировали с различными количествами антитела и через 10 минут инкубировали с первичными NK-клетками человека в качестве эффекторных клеток при соотношении мишень:эффектор 9:1. Антителозависимое уничтожение клеток измеряли с использованием анализа высвобождения LDH (лактатдегидрогеназа) (набор для обнаружения цитотоксичности (LDH) (Roche; кат. № 11644793001)) после 4 часов инкубации при 37 °C.
Макрофагальный или дендритный клеточно-опосредованный антителозависимый клеточный фагоцитоз (ADCP) также может способствовать контролю вируса путем очистки инфицированных клеток и потенциальной стимуляции Т-клеточного ответа презентацией вирусного антигена. ADCP тестировали с использованием мононуклеарных клеток периферической крови в качестве фагоцитов и ExpiCHO, трансфицированных S-белком SARS-CoV-2, флуоресцентно меченным набором флуоресцентных клеточных линкеров PKH67 (Sigma Aldrich, кат. № MINI67) в качестве клеток-мишеней. Клетки-мишени инкубировали с различными количествами антител в течение 10 минут с последующей инкубацией с PBMC человека, выделенными из здоровых доноров, которые были флуоресцентно мечены Cell Trace Violet (Invitrogen, кат. № C34557) при соотношении эффектор:мишень 20:1. После инкубации в течение ночи при 37 °С клетки окрашивали антителом против CD14-APC человека (BD Pharmingen, кат. № 561708, клон M5E2) для окрашивания фагоцитарных клеток. Антитело-опосредованный фагоцитоз определяли с помощью проточной цитометрии, измеряя % моноцитов, которые были положительными на флуоресценцию PKH67.
Были протестированы антитела S309 (VH SEQ ID NO:105; VL SEQ ID NO:168), S304, S306, S315, S230 и комбинация S309 и S304.
На фиг. 17A показана функция ADCC антител с использованием первичных NK-эффекторных клеток и экспрессирующих S-белок SARS-CoV-2 ExpiCHO в качестве клеток-мишеней. Значения показывают среднее значение ±SD (стандартное отклонение) повторных измерений. На фиг. 17B показана функция ADCP антител с использованием PBMC в качестве фагоцитарных клеток и экспрессирующих S-белок ExpiCHO, меченных PKF67, в качестве клеток-мишеней. Значения показывают среднее значение ±SD (стандартное отклонение) повторных измерений.
Варианты Fc S309 тестировали на ADCC. S309-LS включает мутации Fc M428L и N434S. S309-GRLR включает мутацию Fc G236R/L328R, которая демонстрирует минимальное связывание с FcγR. S309-LS-GAALIE включает мутации Fc MLNS и GAALIE (G236A/A330L/I332E). Результаты приведены на фиг. 45.
Антитела S303, S304, S306, S309, S315 и комбинацию S309 и S315 анализировали на функцию ADCC и ADCP. На фиг. 24A показана ADCC антител с использованием первичных NK-эффекторных клеток и экспрессирующих S-белок SARS-CoV- или SARS-CoV-2 ExpiCHO в качестве клеток-мишеней. График на фиг. 24A иллюстрирует процент уничтожения, определенный для одного репрезентативного донора, гомозиготного по высокоаффинному FcγRIIIa (значения показывают среднее ±SD). На фиг. 24В показана площадь под кривой (AUC) для ответов клеток доноров, гомозиготных по высокоаффинному варианту FcγRIIIa 158V (VV), по сравнению с клетками доноров, гетерозиготными по 158V (FV) или гомозиготными по низкоаффинному варианту 158F (FF) (среднее ±SD). На фиг. 25A показан ADCP с использованием PBMC в качестве фагоцитарных клеток и экспрессирующих S-белок SARS-CoV-2 ExpiCHO, меченых PKH67 в качестве клеток-мишеней для одного репрезентативного донора. % ADCP указывает на процент моноцитов, положительных на PKH67. На фиг. 25B показана площадь под кривой (AUC) на ответы нескольких доноров.
Пример 15
Реактивность антител к клеточному лизату клеток, инфицированных SARS-CoV-2
Измеряли реактивность антител S304, S306, S309 и S310 против клеточного лизата клеток VeroE6, инфицированных SARS-CoV-2. На фиг. 21A показана реактивность антител, измеренная с помощью непрямого ELISA S-белка против T×100-экстрагированного лизата клеток VeroE6, инфицированных SARS-CoV-2. На фиг. 21В показана реактивность антител, измеренная с помощью непрямого ELISA S-белка против экстрагированного SDS (денатурированного) лизата клеток VeroE6, инфицированных SARS-CoV-2. На фигуре 21C показана реактивность сыворотки выздоравливающего человека от SARS-CoV-1, измеренная с помощью непрямого ELISA S-белка против экстрагированного T×100 или SDS лизата клеток VeroE6, инфицированных SARS-CoV-2.
Пример 16
Нейтрализация инфекции SARS-CoV-2 с помощью антител S304 и S309 в отдельности или в комбинации
Нейтрализацию инфекции SARS-CoV-2 с помощью моноклональных антител S304 и S309 оценивали с помощью анализа живого вируса SARS-CoV-2. Анализ нейтрализации живого вируса количественно определяет количество инфицированных клеток путем окрашивания вирусного нуклеопротеина (NP) с NP-специфической поликлональной сывороткой кролика. Ингибирование оценивали путем измерения экспрессии NP через 24 и 45 часов после инфицирования. Иммуноферментный анализ (EIA) использовали для количественного определения уровня инфекции для каждого тестируемого разведения антител.
Данные показаны на фиг. 22A-22D. Нейтрализацию проводили в течение одного часа при комнатной температуре при указанных концентрациях антител с использованием клеток Vero E6 в монослое в 96-луночных планшетах. Лунки инфицировали 100 TCID50 вируса. Через 24 или 45 часов монослои фиксировали и окрашивали для ингибирования экспрессии NP. В сочетании S304 и S309 демонстрировали синергетическое усиление нейтрализации.
Пример 17
Получение варианта rIgG антител S309
Рекомбинантные антитела IgG1 получали с использованием последовательностей VH и VL антитела S309. В этом примере антитела называют "S309-11", "S309-12", "S309-13", "S309-14" и "S309-15", соответственно.
"S309-11" содержит последовательность VH дикого типа (SEQ ID NO: 105) и последовательность VL дикого типа (SEQ ID NO: 168) S309. "S309-12" содержит мутацию N55Q в CDRH2, обеспечивающую последовательность варианта VH (SEQ ID NO: 113) и последовательность VL дикого типа (SEQ ID NO: 168) S309. "S309-13" содержит мутацию W50F в VH (SEQ ID NO: 129) и последовательность VL дикого типа (SEQ ID NO: 168) S309. "S309-14" содержит последовательность варианта W105F VH (SEQ ID NO: 119) и последовательность VL дикого типа (SEQ ID NO: 168) S309. "S309-15" содержит вариант W50F/G56A/W105F VH (SEQ ID NO: 172) и последовательность VL дикого типа S309 (SEQ ID NO: 168). Рекомбинантное антитело S309 (S309-11) и каждый из четырех вариантов S309-12 - S309-15 получали путем временной трансфекции и экспрессии плазмидного вектора, кодирующего рекомбинантное антитело, в клетках HD 293F (GenScript). Плазмидный вектор, кодирующий антитела S309, также кодирует сигнальный пептид, как указано в SEQ ID NO:252. Этот сигнальный пептид обеспечивал превосходную продукцию антител по сравнению с другими протестированными сигнальными пептидами. Данные не показаны. Клетки собирали на 4 день, а экспрессию IgG подтверждали с помощью вестерн-блоттинга и анализа титра белка А.
Пример 18
Связывание S309 rIgG и вариантов с RBD SARS-CoV-2
Связывание рекомбинантного моноклонального антитела S309 и четырех вариантов S309, описанных в Примере 17 (S309-12 - S309-15), с RBD измеряли с использованием поверхностного плазмонного резонанса (SPR). Эксперименты SPR проводили с использованием прибора Biacore T200 с использованием кинетического подхода с одним циклом. Антитело, экспрессируемое как IgG, захватывали на поверхности и вводили возрастающие концентрации очищенного RBD SARS-CoV-2 гликозилированной или дегликозилированной формы. SPR проводили с использованием сенсорного чипа с ковалентно иммобилизованным Fc против человека (GE). Использовали буфер 10 мМ HEPES с pH 7,4, 150 мМ NaCl, 3 мМ EDTA и 0,05% моющего средства P20. Анализы проводили при 25 °C. Рекомбинантные антитела разбавляли из супернатанта до приблизительно 2 мкг/мл. Концентрации RBD составляли 0,8 нМ, 3,1 нМ, 12,5 нМ, 50 нМ и/или 200 нМ. Гликозилированную RBD получали путем экспрессии в клетках HEK293 и очищали с использованием одностадийной Ni-аффинной очистки. Дегликозилированный RBD получали путем экспрессии полученной внутренними силами в клетках Expi293, выращенных в присутствии кифунензина, очистки с использованием одностадийной Ni-аффинной очистки и обработки эндогликозидазой H. Кинетические анализы с одним циклом проводили с 3-минутными инъекциями и 20-минутными периодами диссоциации. Кинетику ассоциации и диссоциации контролировали и подгоняли к модели связывания для определения аффинности. Результаты показаны на фиг. 30A-30F и в таблице 8.
Связывание с дегликозилированным RBD измеряли в двух разных анализах SPR с использованием разных параметров. В эксперименте 1 использовали 10-минутные инъекции и серию концентраций RBD из 4-кратных разведений от 100 нМ. В эксперименте 2 использовали 3-минутные инъекции и серию концентраций 4-кратных разведений от 200 нМ, как описано выше. Результаты приведены в таблице 9. Результаты эксперимента 1 для S309-15 также показаны на фиг. 30F, два верхних графика.
Связывание рекомбинантного антитела S309 и четырех сконструированных вариантов с RBD измеряли с помощью поверхностного плазмонного резонанса (SPR) с использованием той же процедуры, описанной выше, за исключением использования очищенных рекомбинантных антител, а не супернатанта клеточной культуры. Результаты показаны в таблице 10.
Пример 19
Нейтрализация инфекции SARS-CoV-2 с помощью антител S309
Нейтрализующую активность S309 и четырех сконструированных вариантов S309, описанных в примерах 17 и 18 ("S309-12" - "S309-15"), определяли с использованием системы репортерного псевдотипирования люциферазы на основе VSV (Kerafast). Псевдотипированные частицы VSV и антитело смешивали в DMEM и оставляли для инкубации в течение 30 минут при 37 °С. Затем инфицирующей смеси давали инкубироваться с клетками Vero E6 в течение 1 часа при 37 °С с последующим добавлением DMEM с Pen-Strep и 10% FBS (инфицирующая смесь не удалялась). Клетки инкубировали при 37 °С в течение 18-24 часов. Люциферазу измеряли с помощью планшетного ридера Ensight (Perkin Elmer) после добавления реагента Bio-Glo (Promega). Результаты приведены на фиг. 28. На фиг. 28 варианты 11 - 15 соответствуют S309-11 - S309-15 соответственно. Расчетные значения EC50, основанные на этом эксперименте, приведены в таблице 11.
Пример 20
Антителозависимая активация FcγRIIIa или FcγRIIa человека
Исследовали антителозависимую активацию FcγRIIIa или FcγRIIa человека. Клетки ExpiCHO временно трансфицировали SARS-CoV-2 (BetaCoV/Wuhan-Hu-1/2019) и инкубировали с титрованными концентрациями антитела в течение 10 минут. Затем клетки ExpiCHO инкубировали с клетками Jurkat, экспрессирующими FcγRIIIa или FcγRIIa на своей поверхности, и стабильно трансфицировали геном люциферазы, управляемым NFAT (Promega, кат. № G9798 и G7018) при соотношении эффектор-мишень 6:1 для FcγRIIIa и 5:1 для FcγRIIa. Активация FcγR человека в этом биологическом анализе приводит к NFAT-опосредованной экспрессии репортерного гена люциферазы. Люминесценцию измеряли после 21 часа инкубации при 37 °С с 5% CO2, используя реагент для анализа люциферазы Bio-Glo-TM в соответствии с инструкциями производителя. Были проанализированы антитела S303, S304, S306, S309, S315 и комбинация S309 и S315, а также сравнительное антитело S230. Результаты показаны на фиг. 31 и 32.
Пример 21
Анализ последовательностей S-гликопротеина SARS-CoV-2
Анализ последовательностей S-гликопротеина 2229 изолятов SARS-CoV-2 показал, что на S-эктодомене SARS-CoV-2 произошло несколько мутаций с переменной частотой. На фиг. 35A показаны варианты спайк-белка, встречающиеся с частотой n более 1 в виде сфер, отображаемых в закрытой и открытой форме полного тримерного спайк-эктодомена. RBD и другие домены спайк-белка показаны как указано. Показано 40 мутаций (из 2229 в общей сложности). Из-за отсутствия детализации в структурах PDB в RBD выделен только остаток 367 (n представляет собой 8), а остатки 476 (n представляет собой 7) и 483 (n представляет собой 17) не выделены. На фиг. 35B показано распространение вариантов спайк-гликопротеинов по аминокислотам. Каждая точка является отдельным вариантом. Показаны местоположения домена A и RBD. Варианты, соответствующие пороговому значению частоты 0,1%, являются такими, как указано.
Дальнейший анализ последовательностей S-гликопротеина проводили с использованием 11 839 изолятов SARS-CoV-2. На фиг. 43 показаны варианты, поддерживаемые по меньшей мере двумя последовательностями (распространенность более 0,01%), визуализируемыми в виде указанных сфер, отображаемых в закрытой (слева) и открытой (справа) форме полного тримерного эктодомена спайка. Каждая точка является отдельным вариантом. На фиг. 43 показаны варианты спайк-белка, подтверждаемые по меньшей мере двумя последовательностями, как указано, сферами, отображенными в закрытой (слева) и открытой (справа) форме полного тримерного спайк-эктодомена. RBD и другие домены спайк-белка показаны как указано. Показаны 171 вариант (из 11 839 проанализированных последовательностей спайк-белка). Варианты помечаются, если их распространенность превышает 1% (только D614G) или если они находятся в пределах RBD. Также указывается местоположение консервации N343.
Пример 22
Конкуренция антитела S309 с антителами, выделенными от пациентов с SARS-CoV-2
Моноклональные антитела человека, выделенные у пациентов, которые выздоровели от инфекции SARS-CoV-2, тестировали на перекрывающиеся сайты связывания RBD с антителом S309 (VH: SEQ ID NO:105; VL: SEQ ID NO:113). Анализы конкуренции проводили с использованием Octet (прибор: Octet Red96, ForteBio). Анти-HIS сенсоры (BIOSENSOR ANTI-PENTA-HIS (HIS1K)1*1ST) использовали для иммобилизации полученного собственными силами His-меченого RBD SARS-CoV-2 (остатки 331-550 спайк-белка из BetaCoV/Wuhan-Hu-1/2019, кат. № MN908947) в концентрации 3 мкг/мл. Антитела ассоциировали в течение 6 мин при 15 мкг/мл. Все белки разбавляли в кинетическом буфере (KB). Затем конкурирующие антитела ассоциировали в той же концентрации в течение дополнительных 6 минут. Было показано, что два антитела конкурируют с S309 за связывание с RBD, но, в отличие от S309, они не нейтрализуют SARS-CoV-2. Данные не показаны.
Пример 23
Выбор устойчивости к SARS-CoV-2 против моноклонального антитела S309-12-MLNS
Для изучения отбора устойчивости, SARS CoV-2 пассировали в течение более одного месяца в присутствии клеток Vero E6 и фиксированных концентраций антитела S309 N55Q MLNS GAALIE (VH с SEQ ID NO:113 и VL с SEQ ID NO:168, с мутациями G236A, A330L, I332E, M428L и N434S в Fc). Экспериментальная схема проиллюстрирована на фиг. 44А. Детали инфекции и непрерывного культивирования вирусов обобщены на фиг. 44В. Цитопатогенный эффект (CPE) оценивали путем визуального осмотра планшетов. Даже при отсутствии CPE титры вирусов оценивали с помощью анализа на бляшкообразование с наложением метилцеллюлозы. Результаты приведены на фиг. 44C. Признаков вирусного прорыва в лунках, обработанных антителами, не наблюдалось даже при минимальной тестируемой концентрации антител. Данные являются репрезентативными для лунок в трех повторностях.
Пример 24
Нейтрализация инфекции SARS-CoV-2 клеток легких человека Calu-3 с помощью антитела S309
Антитело S309 N55Q MLNS (VH с SEQ ID NO:113 и VL с SEQ ID NO:168, с мутациями M428L и N434S в Fc) тестировали на его способность нейтрализовать живую инфекцию SARS-CoV-2 клеток легких человека Calu-3 (которые являются положительными для трансмембранной протеазы TMPRSS2) и клеток VeroE6 с использованием анализа нанолюциферазы. Результаты, включая рассчитанные значения IC50, показаны на фиг. 46.
Пример 25
Нейтрализация инфекции SARS-CoV-2 с помощью антитела S309
Антитело S309 тестировали на его способность нейтрализовать живую инфекцию вируса SARS-CoV-2 с помощью анализа нанолюциферазы и анализа IFA. Вкратце, клетки Vero E6 инфицировали живым вирусом люциферазы SARS-CoV-2 в течение шести часов. Данные собирали с использованием трех различных концентраций антител: 1, 0,1 и 0,01 MOI. Результаты анализа нанолюциферазы показаны на фиг. 47. Результаты анализа IFA показаны на фиг. 48A (репрезентативные лунки, подсчитанные в IFA) и 48B (количественные данные с использованием цитирования 5). Рассчитанные значения IC50 для каждого MOI показаны в полях под графиком на фиг. 47 и 48B. Примечательно, что в данном формате инфекции не наблюдалось скоплений инфекции (или очагов).
Пример 26
Нейтрализация живой инфекции SARS-CoV-2 с помощью антител S309 N55Q MLNS и S309 N55Q MLNS GAALIE
Антитела S309 N55Q MLNS (также называемые в настоящем документе S309 N55Q LS, содержащие мутации Fc M428L/N434S) и S309 N55Q MLNS GAALIE (также называемые в настоящем документе S309 N55Q LS GAALIE, содержащие мутации Fc G236A, A330L, I332E, M428L и N434S) анализировали на способность нейтрализовать инфекцию живым вирусом SARS-CoV-2. Каждый из S309 N55Q MLNS и S309 N55Q MLNS GAALIE содержит VH, имеющий аминокислотную последовательность, представленную в SEQ ID NO:113, и VL, имеющий аминокислотную последовательность, представленную в SEQ ID NO:168. Результаты приведены на фиг. 49. Рассчитанная EC50 для S309 N55Q MLNS составляла 100,1 нг/мл. Рассчитанная EC50 для S309 N55Q MLNS GAALIE составляла 78,3 нг/мл.
Пример 27
Нейтрализация вируса, псевдотипированного SARS-CoV-2 с помощью антител S309 N55Q MLNS и S309 N55Q MLNS GAALIE
Протестировали нейтрализацию вируса, псевдотипированного SARS-CoV-2 с помощью антител S309 N55Q MLNS (также называемыми в настоящем документе S309 N55Q LS) и S309 N55Q MLNS GAALIE (также называемым в настоящем документе S309 N55Q MLNS GAALIE). Каждый из S309 N55Q MLNS и S309 N55Q MLNS GAALIE содержит VH, имеющий аминокислотную последовательность, представленную в SEQ ID NO:113, и VL, имеющий аминокислотную последовательность, представленную в SEQ ID NO:168. Псевдотипированный вирус представлял собой VSV, псевдотипированый спайк-белком SARS-CoV-2. Результаты показаны на фиг. 50A (S309 N55Q MLNS) и фиг. 50B (S309 N55Q MLNS GAALIE). Рассчитанное значение EC50 для S309 N55Q MLNS составляло 24,06 нг/мл. Рассчитанное значение EC50 для S309 N55Q MLNS GAALIE составило 22,09 нг/мл.
Пример 28
Связывание антител S309 N55Q MLNS и S309 N55Q MLNS GAALIE с RBD SARS-CoV-2
Связывание антител S309 N55Q MLNS и S309 N55Q MLNS GAALIE с RBD SARS-CoV-2 измеряли с помощью поверхностного плазмонного резонанса (SPR). Каждый из S309 N55Q MLNS и S309 N55Q MLNS GAALIE содержит VH, имеющий аминокислотную последовательность, представленную в SEQ ID NO:113, и VL, имеющий аминокислотную последовательность, представленную в SEQ ID NO:168. Результаты показаны на фиг. 51A (S309 N55Q MLNS) и фиг. 51B (S309 N55Q MLNS GAALIE).
Пример 29
Связывание антител S309 N55Q MLNS и S309 N55Q MLNS GAALIE со спайк-белком SARS-CoV-2
Связывание антител S309 N55Q MLNS (также называемых в настоящем документе S309 N55Q LS) и S309 N55Q MLNS GAALIE (также называемых в настоящем документе S309 N55Q LS GAALIE) с SARS-CoV-2, со спайк-белком SARS-CoV-2 измеряли с помощью проточной цитометрии. Каждый из S309 N55Q MLNS и S309 N55Q MLNS GAALIE содержит VH, имеющий последовательность, представленную в SEQ ID NO:113, и VL, имеющий последовательность, представленную в SEQ ID NO:168. Результаты показаны на фиг. 51A (S309 N55Q MLNS) и фиг. 51B (S309 N55Q MLNS GAALIE). Данные выражены в виде процента клеток, идентифицированных как положительные на связывание антител.
Пример 30
Связывание антител S309 N55Q MLNS и S309 N55Q MLNS GAALIE с рецепторами Fcγ человека
Связывание антител S309 N55Q MLNS и S309 N55Q MLNS GAALIE с рецепторами Fcγ человека анализировали с использованием SPR. Измеряли связывание с FcγRIIa (как низкоаффинными аллелями R131, так и высокоаффинными аллелями H131), FcγRIIIa (как низкоаффинными аллелями F158, так и высокоаффинными аллелями V158) и FCγRIIb. Каждый из S309 N55Q MLNS и S309 N55Q MLNS GAALIE содержит VH, имеющий последовательность, представленную в SEQ ID NO:113, и VL, имеющий последовательность, представленную в SEQ ID NO:168.
Реагент Biotin CAPture (модифицированный стрептавидин) вводили во все проточные ячейки чипа сенсора CAP, закрепленного в Biacore T200 (Cytiva). Биотинилированные Fc-рецепторы в концентрации 1 мкг/мл вводили через одну проточную кювету со скоростью 10 мкл/мин в течение 60 секунд (один рецептор на проточную кювету), при этом одна проточная кювета резервировалась в качестве эталонной поверхности. Антитело в концентрации 100 мкг/мл (разведенное в HBS-EP+) вводили во все проточные ячейки в течение 200 секунд с использованием скорости потока 30 мкл/мин, и проводили мониторинг ассоциации. Диссоциацию контролировали еще в течение 200 секунд после инъекции. Данные собирали при 10 Гц. После каждого измерения связывания вводили реагент CAP Regeneration для подготовки поверхности к новому циклу. Эксперименты проводили при 25 °C, образцы перед инъекцией выдерживали при 15 °C в приборе. Результаты показаны на фиг. 53 (где мутация MLNS обозначена как "LS" в ключе фигуры).
Пример 31
Связывание антител S309 MLNS, S309 N55Q MLNS и S309 N55Q MLNS GAALIE с компонентом комплемента C1q
Связывание антител S309 MLNS (также называемых в настоящем документе S309 LS), S309 N55Q MLNS (также называемых в настоящем документе S309 N55Q LS) и S309 N55Q MLNS GAALIE (также называемых в настоящем документе S309 N55Q LS GAALIE) с компонентом комплемента C1q измеряли с помощью биослойной интерферометрии (BLI) на приборе Octet. S309 MLNS содержит VH, имеющий последовательность, представленную в SEQ ID NO:105, и VL, имеющую последовательность, представленную в SEQ ID NO:168. Каждый из S309 N55Q MLNS и S309 N55Q MLNS GAALIE содержит VH, имеющий последовательность, представленную в SEQ ID NO:113, и VL, имеющий последовательность, представленную в SEQ ID NO:168.
Античеловеческие Fab (CH1-специфичные) сенсоры использовали для захвата антитела при 10 мкг/мл в течение 10 минут. Затем сенсоры, нагруженные IgG, подвергали воздействию кинетического буфера, содержащего 3 мкг/мл очищенного человеческого C1q, в течение 4 минут с последующей стадией диссоциации в том же буфере в течение дополнительных 4 минут. Профили ассоциации и диссоциации измеряли в режиме реального времени как изменения интерференционной картины. Результаты приведены на фиг. 54.
Пример 32
Активация Fc гамма-рецепторов человека in vitro антителами S309 MLNS, S309 N55Q MLNS и S309 N55Q MLNS GAALIE
Способность антител S309 MLNS, S309 N55Q MLNS и S309 N55Q MLNS GAALIE вызывать антителозависимую активацию рецепторов Fcγ человека анализировали in vitro. S309 MLNS содержит VH, имеющий последовательность, представленную в SEQ ID NO:105, и VL, имеющий последовательность, представленную в SEQ ID NO:168. Каждый из S309 N55Q MLNS и S309 N55Q MLNS GAALIE содержит VH, имеющий последовательность, представленную в SEQ ID NO:113, и VL, имеющий последовательность, представленную в SEQ ID NO:168.
Каждый из S309 MLNS (также называемый в настоящем документе S309 LS), S309 N55Q MLNS (также называемый в настоящем документе S309 N55Q LS), S309 N55Q MLNS GAALIE (также называемый в настоящем документе S309 N55Q LS GAALIE) и контрольное антитело S309-GRLR серийно разводили в 6 раз в буфере для анализа от 10 000 нг/мл до 0,006 нг/мл. Девятиточечные серийные разведения антитела инкубировали с 12 500 (для FcγRIIIa и FcγRIIb) или 10000 (для FcγRIIa) клеток CHO со спайк-белком CoV-2 на 96-луночную планшетную лунку в белом планшете с плоским дном в течение 15 минут при комнатной температуре. Эффекторные клетки Jurkat, экспрессирующие указанные FcγR и стабильно трансфицированные геном люциферазы, управляемым NFAT, размораживали, разбавляли в буфере для анализа и добавляли в планшет при соотношении эффекторных и целевых клеток 6:1 для FcRγIIIa и FcγRIIb или 5:1 для FcγRIIa. Контрольные лунки были включены для измерения независимой от антитела активации (содержащей клетки-мишени и эффекторные клетки, но не содержащей антитела) и фоновой люминесценции планшета (лунки, содержащие только аналитический буфер). Планшеты инкубировали в течение 18 часов при 37 °C с 5% CO2. Активация FcγR человека в этом биологическом анализе приводит к NFAT-опосредованной экспрессии репортерного гена люциферазы. Люминесценцию измеряли с помощью люминометра после добавления реагента для анализа люциферазы Bio-Glo™ в соответствии с инструкциями производителя. Результаты приведены на фиг. 55.
Пример 33
Эффекторная функция антител S309 MLNS, S309 N55Q MLNS и S309 N55Q MLNS GAALIE
Антитела S309 MLNS (также называемые в настоящем документе как S309 LS), S309 N55Q MLNS (также называемые в настоящем документе как S309 N55Q LS) и S309 N55Q MLNS GAALIE (также называемые в настоящем документе как S309 N55Q LS GAALIE) анализировали на их способность стимулировать антитело-зависимую от NK-клеток клеточно-опосредованную цитотоксичность (ADCC) и моноцитарно-опосредованный антитело-зависимый клеточный фагоцитоз (ADCP) против клеток, экспрессирующих спайк-белок CoV-2.
S309 MLNS содержит VH, имеющий последовательность, представленную в SEQ ID NO:105, и VL, имеющую последовательность, представленную в SEQ ID NO:168. Каждый из S309 N55Q MLNS и S309 N55Q MLNS GAALIE содержит VH, имеющий последовательность, представленную в SEQ ID NO:113, и VL, имеющий последовательность, представленную в SEQ ID NO:168.
ADCC измеряли in vitro путем экспонирования свежевыделенных NK-клеток человека от двух генотипированных доноров, экспрессирующих гомозиготный низкоаффинный (F/F158) или высокоаффинный (V/V158) FcγRIIIa, к антителу, предварительно инкубированному с клетками со спайк-белком CHO-CoV-2, и измерения высвобождения LDH в качестве считывания в соответствии с инструкциями производителя (набор для обнаружения цитотоксичности (LDH), Roche) после 4 часов инкубации при 37 °C. Вкратце, планшеты центрифугировали в течение 4 минут при 400 x g, и 35 мкл супернатанта переносили в плоский 384-луночный планшет. Готовили реагент LDH и добавляли 35 мкл в каждую лунку. Используя кинетический протокол, поглощение при 490 нм и 650 нм измеряли один раз каждые 2 минуты в течение 8 минут, и в качестве результата использовали наклон кинетической кривой. Процент специфического лизиса определяли с помощью следующей формулы: (специфическое высвобождение - спонтанное высвобождение)/(максимальное высвобождение - спонтанное высвобождение) ×100. Результаты приведены на фиг. 56.
Способность антител S309 MLNS, S309 N55Q MLNS, S309 N55Q MLNS GAALIE и контрольного антитела S309-GRLR стимулировать ADCP первичными моноцитами CD14+ измеряли in vitro путем воздействия свежевыделенными PBMC человека (мечеными cell trace violet) на CHO клетки, экспрессирующие спайк-белок CoV-2 (мечеными набором флуоресцентных клеточных линкеров PKH67 (Sigma Aldrich)), которые предварительно инкубировали с антителом. Последовательные разведения mAb (серийно разведенных в 5 раз от 5000 нг/мл до 0,32 нг/мл в RPMI-1640 (PPMI - Онкологический институт имени Розуэлла Парка) + L-глутамин с добавлением 10% Hyclone FBS + 2x анти-анти (антибиотик-антимикотик)) инкубировали с 10 000 клеток CHO со спайк-белком CoV-2 на лунку 96-луночного полипропиленового планшета в течение 10 минут. Первичные PBMC были флуоресцентно мечены Cell Trace Violet в соответствии с инструкциями производителя. Затем смеси клеток-мишеней и антител инкубировали с мечеными PBMC в соотношении эффектор/мишень 16:1. Активность ADCP измеряли после инкубации в течение ночи путем мечения популяции моноцитов для CD14 и измерения процента Cell trace violet+ PKH67+ среди моноцитов CD14+ с помощью проточной цитометрии. Результаты приведены на фиг. 57.
Пример 34
Влияние антитела S309 на слияние клеток, опосредованное спайк-белком SARS-CoV-2
Влияние антитела S309 (VH: SEQ ID NO:105; VL: SEQ ID NO:168) на слияние, опосредованное спайк-белком SARS-CoV-2, тестировали с использованием клеток, сконструированных для сверхэкспрессии спайк-белка на поверхности клетки. Добавление S309 к этим клеточным культурам ингибировало слияние клетка-клетка. Результаты показаны на фиг. 58A (микрофотографии) и 58B (количественные данные).
Пример 35
Влияние антител S309 N55Q MLNS и S309 N55Q MLNS GAALIE на репликацию SARS-CoV-2
Влияние антител S309 N55Q MLNS и S309 N55Q MLNS GAALIE на репликацию SARS-CoV-2 испытывали в клетках VeroE6, PBMC и дендритных клетках. Каждый из S309 N55Q MLNS и S309 N55Q MLNS GAALIE содержит VH, имеющий последовательность, представленную в SEQ ID NO:113, и VL, имеющий последовательность, представленную в SEQ ID NO:168.
Вирус SARS-CoV-2 инкубировали в течение одного часа с S309 N55Q MLNS или S309 N55Q MLNS GAALIE. Затем смесь вирус/антитело добавляли к высеянным на планшет клеткам VeroE6, PBMC или дендритным клеткам, полученным из моноцитов (MoDC). После инкубации клеток со смесью вирус/антитело в течение одного часа при 37 °С, клетки промывали и инкубировали в течение еще 72 часов в свежей среде. Затем супернатант из культивируемых клеток анализировали на бляшкообразующие единицы (FFU). Супернатант разбавляли 1:5 и добавляли к клеткам VeroE6. Через один час при 37 °С клетки VeroE6 накладывали метилцеллюлозой. Через 24 часа дальнейшей инкубации культуры клеток VeroE6 окрашивали на нуклеопротеин SARS-CoV-2. Результаты приведены на фиг. 59. Данные для антитела S309 N55Q MLNS показаны на верхнем графике. Данные для антитела S309 N55Q MLNS GAALIE показаны на нижнем графике. Эти 72-часовые данные репликации являются репрезентативными для результатов через 24 и 48 часов.
Пример 36
Материалы и методы
Скрининг на основе проточной цитометрии для связывания с S-белком CoV, экспрессируемым на клетках млекопитающих
Клетки ExpiCHO трансфицировали S-белком SARS-CoV-2, SARS-CoV и MERS-CoV или пустой плазмидой в качестве отрицательного контроля. Затем моноклональные антитела тестировали с помощью проточной цитометрии в концентрации 10 мкг/мл на их способность окрашивать клетки ExpiCHO, экспрессирующие S-белок 2019-nCoV, SARS-CoV, MERS-CoV или имитационная клетка трансфектант.
Временная экспрессия рекомбинантного белка SARS-CoV-2
Полноразмерный ген S штамма SARS-CoV-2 (2019-nCoV-S) изолята BetaCoV/Wuhan-Hu-1/2019 (кат. № MN908947) был кодон-оптимизирован для экспрессии клеток человека и клонирован в вектор экспрессии phCMV1 (Genlantis). Клетки Expi-CHO временно трансфицировали phCMV1-SARS-CoV-2-S, phCMV1-MERS-CoV-S (London1/2012), SARS-spike_pcDNA.3 (штамм SARS) или пустой phCMV1 (имитация) с использованием Expifectamine CHO Enhancer. Через два дня после трансфекции клетки собирали, фиксировали или фиксировали и пермеабилизировали сапонином для иммуноокрашивания панелью моноклональных антител, реагирующих на рецептор-связывающий домен (RBD) SARS-CoV. Для обнаружения использовали меченное Alexa647 вторичное антитело против Fc IgG человека. Связывание антитела с трансфицированными клетками анализировали с помощью проточной цитометрии с использованием клеточного анализатора ZE5 (Biorard) и программного обеспечения FlowJo (TreeStar). Положительное связывание определяли дифференциальным окрашиванием CoV-S трансфектантов по сравнению с имитационными трансфектантами.
Эксперименты по конкурированию с использованием Octet (BLI, биослойная интерферометрия)
Если в настоящем документе не указано иное, для иммобилизации S1 субъединцы белка SARS-CoV (Sino Biological Europe GmbH) использовали анти-His сенсоры (BIOSENSOR ANTI-PENTA-HIS (HIS1K)). Сенсоры гидратировали в течение 10 мин кинетическим буфером (KB; 0,01% BSA без эндотоксинов, 0,002^ Tween-20, 0,005% NaN3 в PBS). Затем субъединицу S1 белка SARS-CoV загружали в течение 8 мин в концентрации 10 мкг/мл в KB. Антитела ассоциировали в течение 6 мин при 15 мкг/мл для полноразмерных mAb nCoV-10 и nCov-6 mAb или 5 мкг/мл для Fab nCoV-4 и в последующем эксперименте, включающем nCoV-1 все при 10 мкг/мл. Затем конкурирующие антитела ассоциировали в той же концентрации в течение дополнительных 6 минут.
Эксперименты по конкурированию с использованием Octet (BLI, биослойная интерферометрия)
Для экспериментов по конкурированию ACE2, ACE2-His (Bio-Techne AG) загружали в течение 30 минут при 5 мкг/мл в KB на анти-HIS биосенсоры (HIS2) (Molecular Devices-ForteBio). SARS-CoV-1 RBD-rabbitFc или SARS-CoV-2 RBD-mouseFc (Sino Biological Europe GmbH) в концентрации 1 мкг/мл ассоциировали в течение 15 минут после предварительной инкубации с антителом или без антитела (30 мкг/мл, 30 минут). Диссоциацию контролировали в течение 5 минут.
Определение аффинности с использованием Octet (BLI, биослойная интерферометрия)
Для определения KD полноразмерных антител использовали биосенсоры белка A (Pall ForteBio) для иммобилизации рекомбинантных антител при 2,7 мкг/мл в течение 1 минуты после этапа гидратации в течение 10 минут с помощью кинетического буфера. Кривые ассоциации регистрировали в течение 5 минут путем инкубации сенсоров, покрытых антителами, с различной концентрацией RBD SARS-CoV-1 (Sino Biological) или RBD SARS-CoV-2 (полученный собственными силами; остатки 331-550 спайк-белка BetaCoV/Wuhan-Hu-1/2019, кат. № MN908947). Самая высокая протестированная концентрация RBD составляла 10 мкг/мл, затем серийно разбавляли 1:2,5 с. Диссоциацию регистрировали в течение 9 минут, перемещая сенсоры в лунки, содержащие KB. Значения KD рассчитывали с использованием глобальной модели соответствия (Octet). Использовали оборудование Octet Red96 (ForteBio).
Для определения KD полноразмерных антител по сравнению с Fab-фрагментами His-меченные RBD SARS-CoV-1 или SARS-CoV-2 загружали в концентрации 3 мкг/мл в KB в течение 15 минут на анти-HIS биосенсоры (HIS2) (Molecular Devices, ForteBio). Ассоциацию полноразмерного антитела и Fab проводили в KB при 15 мкг/мл и 5 мкг/мл соответственно в течение 5 минут. Диссоциацию в KB измеряли в течение 10 минут.
Связывание ELISA
Реактивность mAb с субъединицей S1 спайк-белка SARS-CoV (штамм WH20) определяли с помощью твердофазного иммуноферментного анализа (ELISA). Вкратце, 96-луночные планшеты покрывали 3 мкг/мл субъединицы S1 рекомбинантного спайк-белка SARS-CoV (Sino. Biological). Лунки промывали и блокировали PBS+1%BSA в течение 1 часа при комнатной температуре, а затем инкубировали с серийно разведенными mAb в течение 1 часа при комнатной температуре. Связанные mAb обнаруживали путем инкубации конъюгированных с щелочной фосфатазой козьих антител против IgG человека (Southern Biotechnology: 2040-04) в течение 1 часа при комнатной температуре и проявляли с помощью 1 мг/мл п-нитрофенилфосфатного субстрата в 0,1 М глициновом буфере (pH 10,4) в течение 30 минут при комнатной температуре. Оптическую плотность (OD) измеряли при длине волны 405 нм в ридере ELISA (спектрофотометр Powerwave 340/96, BioTek).
Анализ нейтрализации
Если не указано иное, использовали вирус мышиного лейкоза (MLV), псевдотипированный спайк-белком SARS-CoV-2 (SARS-CoV-2pp) или спайк-белком SARS-CoV-1 (SARS-CoV-1pp). В качестве клеток-мишеней использовали DBT-клетки, стабильно трансфицированные ACE2 (DBT-ACE2). SARS-CoV-2pp или SARS-CoV-1pp активировали трипсином TPCK в концентрации 10 мкг/мл. Активированный SARS-CoV-2pp или SARS-CoV-1pp добавляли к серии разведений антител (начиная с конечной концентрации 50 мкг/мл на антитело, 3-кратное разведение). Клетки DBT-ACE2 добавляли к смесям антитело-вирус и инкубировали в течение 48 часов. Люминесценцию измеряли после аспирации супернатанта клеточной культуры и добавления устойчивого субстрата GLO (Promega).
Если не указано иное, в анализах нейтрализации псевдотипированных частиц используется система псевдотипирования репортеров люциферазы на основе VSV (Kerafast). Псевдотипированные частицы VSV и антитело смешивали в DMEM и оставляли инкубироваться в течение 30 минут при 37 °C. Инфекционную смесь затем инкубировали с клетками Vero E6 в течение 1 часа при 37 °C с последующим добавлением DMEM с Pen-Strep и 10% FBS (инфекционная смесь не удаляется). Клетки инкубировали при 37 °C в течение 18-24 часов. Люциферазу измеряли с помощью планшетного ридера Ensight (Perkin Elmer) после добавления реагента Bio-Glo (Promega).
SPR кинетического подхода с одним циклом
Эксперименты SPR проводили с использованием прибора Biacore T200 с использованием кинетического подхода с одним циклом. S309 IgG захватывали на поверхности и вводили возрастающие концентрации очищенного RBD SARS-CoV-2 гликозилированного или дегликозилированного. Кинетику ассоциации и диссоциации контролировали и подгоняли к модели связывания для определения аффинности.
Экспрессия рекомбинантных антител
Рекомбинантные антитела экспрессировали в клетках ExpiCHO, временно котрансфицированных плазмидами, экспрессирующими тяжелую и легкую цепи, как описано ранее. (Stettler и др. (2016) Specificity, cross-reactivity, and function of antibodies elicited by Zika virus infection. Science, 353(6301), 823-826) Моноклональные антитела S303, S304, S306, S309, S310 и S315 экспрессировали в виде антител rIgG-MLNS. Мутация MLNS обеспечивает более длительный период полувыведения in vivo. (Zalevsky и др. (2010) Enhanced antibody half-life improves in vivo activity. Nature Biotechnology, 28(2), 157-159)
Выравнивание последовательностей
Геномные последовательности SARS-CoV-2 были загружены из GISAID 29 марта 2020 г. с использованием фильтров «полный (более 29 000 п.н. (пар нуклеотидов))» и «исключение с низким охватом». Последовательности летучих мышей и панголина удаляли с получением последовательностей только для человека. ORF (открытая рамка считывания) спайк-белка локализовали путем выполнения выравнивания генома эталонного белка (YP_009724390.1) с GeneWise2. Неполные совпадения и ORF, содержащие вставку/делецию, были сохранены и включены в последующий анализ. Нуклеотидные последовательности транслировали in silico с использованием seqkit. Удаляли последовательности с более чем 10% неопределенных аминокислот (из-за распознавания N оснований). Выравнивание нескольких последовательностей проводили с использованием MAFFT. Варианты определяли путем сравнения выровненных последовательностей (n сосавляет 2229) с эталонной последовательностью с использованием R/Bioconductor Package Biostrings. Аналогичную стратегию использовали для извлечения и трансляции последовательностей спайк-белка из геномов SARS-CoV, полученных из ViPR (критерии поиска: коронавирус, связанный с SARS, полноразмерные геномы, хозяин-человек, депонированный до декабря 2019 года, чтобы исключить SARS-CoV-2, n составляет 53). Исходные последовательности генома SARS-CoV содержали все основные опубликованные штаммы, такие как Urbani, Tor2, TW1, P2, Frankfurt1 и другие. Последовательности панголина, показанные в Tsan-Yuk Lam и др., были получены из GISAID. Последовательности летучих мышей из трех кладов сарбековирусов, как показано в Lu и др. (Lancet 2020), были получены из Genbank. Последовательности циветы и енотовидной собаки аналогичным образом были получены из Genbank.
Пример 37
ACE2-независимый механизм нейтрализации SARS-CoV2 с помощью антитела S309
В следующих экспериментах антитело S309 (VH с SEQ ID NO:105, VL с SEQ ID NO:168) экспрессировали в виде рекомбинантного IgG1 с мутациями Fc M428L и N434S. Исследовали влияние сверхэкспрессии ACE2 на нейтрализацию инфекции спомощью антитела S309. Клетки Vero E6 или Vero E6-TMPRSS2 инфицировали SARS-CoV-2 (изолят USA-WA1/2020) при MOI 0,01 в присутствии S309 (10 мкг/мл). Клетки фиксировали через 24 часа после инфицирования, вирусный нуклеокапсидный белок подвергали иммуноокрашиванию и количественному определению. Окрашивание нуклеокапсида практически отсутствовало в клетках, обработанных антителами. S309 имело IC50 (нг/мл) в клетках Vero E6, составляла 65, и в клетках Vero E6-TMPRSS2, составляла 91 (данные не показаны).
Панель из 7 клеточных линий (HeLa, 293T (wt), Vero E6, Huh7, 293T ACE2, MRC 5-ACE2-TMPRSS2, A549-ACE2-TMPRSS2 клон 5, A549-ACE2-TMPRSS2 клон 10) была инфицирована SARS-CoV-2-Nluc или VSV, псевдотипированным спайк-белком SARS-CoV-2 в присутствии S309. Сигнал люциферазы определяли количественно через 24 часа после инфицирования. Максимальные значения нейтрализации S309 были такими, как показано в таблице 12.
Связывание очищенного, меченного флуоресцентной меткой спайк-белка SARS-CoV-2 с этими клеточными линиями количественно определяли с помощью проточной цитометрии. Клетки HeLa и 239T WT имели самые низкие значения MFI, за которыми следовали клетки Huh7 и VeroE6. Клетки 293T ACE2 (максимальная нейтрализация), MRC 5-ACE2-TMPRSS2 (третье по величине значение нейтрализации), клон 5 A549-ACE2-TMPRSS2 (четвертое по величине значение нейтрализации) и клон 10 A549-ACE2-TMPRSS2 (второе по величине значение нейтрализации) имели более высокие значения MFI. Был определен корреляционный анализ между максимальным потенциалом нейтрализации связывания с добавкой S309; значения корреляции Spearman S309 составили: r составляет -0,94 для обеих вирусных моделей. p составляет 0,017.
Чтобы дополнительно охарактеризовать клеточные линии, чувствительные к SARS-CoV-2, семь клеточных линий, описанных выше, инкубировали с очищенным, флуоресцентно меченным спайк-белком SARS-CoV-2 или белком RBD, и связывание белка количественно определяли с помощью проточной цитометрии. В порядке убывания MFI клеточные линии представляли собой: A549-ACE2-TMPRSS2 клон 10; 293T ACE2; MRC 5-ACE2-TMPRSS2; A549-ACE2-TMPRSS2 клон 5; Vero E6; Huh7; 293T (wt); и HeLa.
Отобранные лектины и опубликованные кандидаты в рецепторы были проверены с использованием клеток HEK293T, инфицированных вирусами VSV, псевдотипированными SARS-CoV-2. ACE2, DC-SIGN, L-SIGN и SIGLEC-1 дали самые высокие сигналы. ACE2 обеспечивал сигнал приблизительно 105 относительных люминесцентных единиц (RLU), а DC-SIGN, SIGLEC-1 и L-SIGN имели сигналы приблизительно 104 RLU. Все остальные протестированные лектины/кандидаты дали сигналы приблизительно 102-103 RLU.
Клетки HEK 293T, HeLa и MRC5 временно трансдуцировали для сверхэкспрессии DC-SIGN, L-SIGN, SIGLEC1 или ACE2 и инфицировали вирусами VSV, псевдотириванными SARS-CoV-2. Неинфицированные клетки и нетрансдуцированные клетки были включены в качестве контролей. В клетках HEK293T ACE2, DC-SIGN, SIGLEC-1 и L-SIGN обеспечивали значительное усиление инфекции. В клетках HeLa и MRC5 только ACE2 усиливал инфекцию.
Стабильные клеточные линии HEK293T, сверхэкспрессирующие DC-SIGN, L-SIGN, SIGLEC-1 или ACE2, инфицировали аутентичным SARS-CoV-2 (MOI 0,1), фиксировали и иммуноокрашивали через 24 часа для нуклеопротеина SARS-CoV-2. В качестве контролей использовали клетки дикого типа (инфицированные и неинфицированные). Усиленное окрашивание наблюдалось в клетках, сверхэкспрессирующих DC-SIGN, L-SIGN или SIGLEC-1, и окрашивание было значительно увеличено в клетках, сверхэкспрессирующих ACE2.
Стабильные клеточные линии инфицировали SARS-CoV-2-Nluc, а уровни люциферазы определяли количественно через 24 часа. В порядке возрастания RLU: неинфицированные (около 102-103 RLU); исходные 293T (около 104 RLU); DC-SIGN (около 105 RLU); L-SIGN (около 105 RLU); SIGLEC-1 (около 105-106 RLU ); ACE2 (более 107 RLU).
Стабильные клеточные линии инкубировали с различными концентрациями анти-SIGLEC1 mAb (клон 7-239) и инфицировали SARS-CoV-2-Nluc. Инфекция в процентах от необработанных клеток оставалась близкой к превышению 100% в клетках 293T, экспрессирующих DC-SIGN, L-SIGN или ACE2, но упала ниже 50% (0,2 мкг/мл анти-SIGLEC) до уровня, близкого к 0 (1 мкг/мл или 5 мкг/мл анти-SIGLEC) в клетках 293T, экспрессирующих SIGLEC-1.
Уровни экспрессии отдельных потенциальных кандидатов корецептора SARS-CoV-2 в одной клетке определяли в различных типах клеток легкого, полученных из Human Lung Cell Atlas (Атлас клеток легких человека) (nature.com/articles/s41586-020-2922-4). DC-SIGN, L-SIGN и SIGLEC-1 экспрессируются в различных типах клеток в легком на уровнях, аналогичных или даже выше, чем ACE2.
Связывание антител, нацеленных на DC-/L-SIGN, DC-SIGN, SIGLEC1 или ACE2 на клетках HEK293T, стабильно сверхэкспрессирующих соответствующий рецептор связывания, анализировали с помощью проточной цитометрии и иммунофлуоресцентного анализа. Клетки HEK 293T, сверхэкспрессирующие соответствующие рецепторы связывания, инфицировали VSV, псевдотипированным спайк-белком дикого типа или мутантным спайк-белком SARS-COV-2 линии B1.1.7. Люминесценцию анализировали на следующий день после инфицирования. Инфекция была усилена в клетках, экспрессирующих рецепторы связывания. Инфекция с помощью VSV, псевдотипированного любым спайк-белком, была аналогичной для каждой испытуемой группы. Клетки, экспрессирующие ACE2, давали самый высокий сигнал люминесценции.
Клетки Vero E6, дифференцированные in vitro moDC или PBMC инфицировали SARS-CoV-2 с MOI 0,01. Через 24 часа после инфицирования клетки фиксировали, иммуноокрашивали на вирусный нуклеокапсидный белок и количественно определяли инфицированные клетки. Только клетки VeroE6 показали инфекцию (около 7% клеток). Супернатант инфицированных клеток брали через 24, 48 и 72 часа и количественно определяли титр инфекционного вируса с помощью анализа FFU на клетках Vero E6.
Были оценены основные типы клеток с обнаруживаемым геномом SARS-CoV-2 в жидкости бронхоальвеолярного лаважа (BALF) и мокроте пациентов с тяжелым течением COVID-19. Был построен график t-SNE, и было определено количество каждого типа клеток SARS-CoV-2+ (всего n составляет 3085 клеток от 8 субъектов в публикации Ren и др. Cell 2021). Типы клеток включали T-клетки, NK-клетки, клетки плазмы, нейтрофилы, макрофаги, реснитчатые клетки, плоскоклеточные и секреторные. Экспрессию ACE2, DC-SIGN, L-SIGN, SIGLEC-1 и их комбинаций оценивали для каждого типа клеток.
Количество транскриптов ACE2, DC-SIGN (CD209), L-SIGN (CLEC4M), SIGLEC1 коррелировали с количеством РНК SARS-CoV-2 в макрофагах и секреторных клетках. Корреляция была основана на подсчетах (до преобразования логарифма), от Ren и др. Cell 2021).
Репрезентативные данные, демонстрирующие экспрессию рецепторов в стабильных клеточных линиях HEK293T, показаны на фиг. 60. Клеточные линии генерировали для сверхэкспрессии DC-SIGN, L-SIGN или ACE2 путем трансдукции клеток HEK293T лентивирусом, кодирующим трансген, и проводили иммунофлуоресцентные анализы для оценки экспрессии трансгена.
Репрезентативные данные, демонстрирующие способность псевдотипированного вируса VSV, экспрессирующего S-белок SARS-CoV-2 с репортером люциферазы, инфицировать клетки HEK293T (с использованием люминесцентного анализа), показаны на фиг. 61; экспрессия DC-SIGN или L-SIGN увеличивала уровни инфекции псевдотипированного вируса более чем в 10 раз по сравнению с инфекцией клеток HEK293T WT, и экспрессия ACE2 увеличивала уровни инфекции пседотипированным вирусом более чем в 100 раз по сравнению с инфекцией клеток HEK293T WT.
Нейтрализующую активность примерного mAb S309 против пседотипированного вируса VSV оценивали в сконструированных клетках HEK293T. Данные показаны на фиг. 62; S309 полностью нейтрализовало инфекцию с помощью DC-SIGN и L-SIGN и, в меньшей степени, ACE2.
Способность живого SARS-CoV-2 с репортером люциферазы инфицировать клетки HEK293T исследовали с помощью люминесцентного анализа. Данные показаны на фиг. 63; экспрессия DC-SIGN или L-SIGN увеличивала уровни инфекции живым вирусом более чем в 3 раза по сравнению с инфекцией клеток HEK293T WT, и экспрессия ACE2 увеличивала уровни инфекции живым вирусом более чем в 100 раз по сравнению с инфекцией клеток HEK293T WT.
Нейтрализующую активность mAb S309 против пседотипированного вируса VSV оценивали в сконструированных клетках HEK293T. Данные показаны на фиг. 64; S309 полностью нейтрализовало инфекцию с помощью DC-SIGN и L-SIGN и, в меньшей степени, нейтрализовало заражение через ACE2.
Были проведены эксперименты для изучения того, может ли антитело S309 нейтрализовать проникновение SARS-CoV-2 через SIGLEC-1. Вкратце, стабильные клеточные линии HEK293T создавали, как описано выше, для сверхэкспрессии DC-SIGN/L-SIGN, DC-SIGN, SIGLEC-1 или ACE2. Данные экспрессии показаны на фиг. 65. Как показано на фиг. 66; экспрессия DC-SIGN, L-SIGN или SIGLEC увеличивала уровни инфекции живым вирусом более чем в 10 раз по сравнению с инфекцией клеток HEK293T WT, и экспрессия ACE2 увеличивала уровни инфекции псевдотипированным вирусом более чем в 100 раз по сравнению с инфекцией клеток HEK293T WT. Как показано на фиг. 67, S309 полностью нейтрализовало инфекцию через DC-SIGN, L-SIGN и SIGLEC-1.
Экспрессию DC-SIGN (CD209) и других белков рецепторов клеточной поверхности, включая SIGLEC-1 и другие SIGLEC, определяли на различных типах клеток. Данные обобщены на фиг. 68A и 68B.
Были проведены дальнейшие эксперименты для исследования функции (функций) DC-SIGN, L-SIGN и SIGLEC-1 при инфекции SARS-CoV-2. В одном наборе экспериментов клетки HEK293T, стабильно экспрессирующие DC-SIGN, L-SIGN, SIGLEC-1 или ACE2, инфицировали живым Nluc SARS-CoV-2 при трех различных множествах инфекций (MOI): 0,01, 0,1 и 1). Инфекцию определяли с использованием относительных люминесцентных единиц и сравнивали с инфекцией в клетках HEK293T (исходных). Данные показаны на фиг. 69. При наименьшем тестируемом MOI наблюдалось увеличение инфекции в клетках, экспрессирующих DC-SIGN, L-SIGN или SIGLEC. На самом высоком тестируемом MOI, инфекция не была дополнительно усилена по сравнению с исходной экспрессией DC-SIGN, L-SIGN или SIGLEC. Эти данные показывают, что тсходные клетки 293T восприимчивы к инфекции SARS-CoV-2 и L-SIGN, DC-SIGN и SIGLEC-1 усиливают уровни инфекции, но не функционируют как первичные рецепторы инфекции.
В другом наборе экспериментов клетки 293T, клетки HeLa и клетки MRC5 временно трансдуцировали лентивирусом, кодирующим DC-SIGN, L-SIGN, SIGLEC-1 или ACE2, и инфицировали псевдотипированным вирусом VSV через три дня после трансдукции. Данные показаны на фиг. 70. В то время как клетки 293T показали низкий уровень восприимчивости (по сравнению с неинфицированными и нетрансдуцированными), клетки HeLa и MRC5 были полностью устойчивы к вирусу. Низкий уровень инфекции в клетках 293T может быть увеличен путем экспрессии L-SIGN, DC-SIGN или SIGLEC-1, что соответствует роли этих белков, а также факторов связывания. Клетки HeLa и MRC5 оставались устойчивыми к инфекции даже после экспрессии L-SIGN, DC-SIGN или SIGLEC-1 и становились восприимчивыми только после экспрессии ACE2. Эти данные показывают, что L-SIGN, DC-SIGN и SIGLEC-1 не являются первичными рецепторами для SARS-CoV-2.
Пример 38
Эффективность in vivo антитела S309
Эффективность S309 исследовали на сирийских хомяках. Эта животная модель представляет собой на сегодняшний день наиболее релевантную модель инфекции SARS-CoV-2, которая не требовала чрезмерной экспрессии in vivo ACE2 для поддержания продуктивной инфекции и заболевания. Профилактическое введение S309 индуцировало дозозависимую защиту от инфекции SARS-CoV-2 и повреждения тканей у хомяков, что продемонстрировали уровни вирусной РНК, вирусная нагрузка, а также гистопатологическая оценка легких (фиг. 73A-7C). Эти данные показывают, что плохая и неполная нейтрализация проникновения S309 in vitro при использовании клеток, сверхэкспрессирующих ACE2, не нарушала in vivo эффективность mAb, не имеющих RBM.
S309, несущее мутацию N297A, имеет сниженную способность запускать эффекторные функции вследствие снижения взаимодействия с рецепторами Fcγ. Это было дополнительно подтверждено снижением связывания варианта S309-N297A с моноцитами хомяка в селезенке. Эффективность in vivo, измеренная с помощью mAb N297A, аналогична или просто немного уступает S309 wt, что указывает на то, что нейтрализующая способность mAb преобладает над его способностью выполнять эффекторную функцию в этих условиях. Концентрация S309 в сыворотке, необходимая для снижения вирусной РНК в легком на 90%, составляла 9 мкг/мл, фиг. 73D.
Пример 39
Активность антител против вариантов SARS-CoV-2
Появился ряд вариантов SARS-CoV-2, при этом в конце 2020 года было зарегистрировано увеличение числа случаев инфицирования вариантами. Мотив связывания рецептора (RBM), по-видимому, является особенно изменчив в отношении к мутации. Заметные появляющиеся варианты наблюдались в Шотландии, Великобритании, Южной Африке, Калифорнии, Колумбусе и у норок в Дании, и сообщалось, что некоторые мутации обеспечивают ускользание от антител или нейтрализацию сыворотки. Эксперименты проводили для оценки способности антител S309 нейтрализовать варианты. S309 N55Q MLNS (VH: SEQ ID NO:113; VL: SEQ ID NO:168; с мутациями Fc M428L и N434S) тестировали против SARS-CoV-2, с панелью из 20 наиболее часто встречающихся мутаций варианта RBD SARS-CoV-2, как определено с помощью считывания последовательности. Антитела REGN10933 и REGN10987 (Hansen и др., Science 369(6506):1010-1014; eabd0827-0810 (2020) и PDB 6XDG (rcsb.org/structure/6XDG)) были включены для сравнения. Результаты обобщены в Таблице 13.
Y означает менее чем трехкратное снижение нейтрализации живого вируса или псевдотипированного вируса;
N означает более чем трехкратное снижение нейтрализации живого вируса или псевдотипированного вируса;
P представляет собой нейтрализацию антителом прогнозируется из-за того, что вариантная аминокислота находится вне эпитопа;
? означает неизвестно.
Y означает менее чем трехкратное снижение нейтрализации живого вируса или псевдотипированного вируса;
N означает более чем трехкратное снижение нейтрализации живого вируса или псевдотипированного вируса;
P представляет собой нейтрализацию антителом прогнозируется из-за того, что вариантная аминокислота находится вне эпитопа;
? означает неизвестно.
Общее количество секвенированных мутантов SARS-CoV-2, которые, как известно, ускользают от антител (по состоянию на 29 января 2021 г.), составляло: S309 N55Q MLNS = 29; REGN10987 = 10425; REGN10933 = 3621.
Связывание антител S309 с вариантом RBD SARS-CoV-2 оценивали с помощью BLI. Оценивали S309 (VH: SEQ ID NO:105; VL: SEQ ID NO:168) с Fc дикого типа и S309 N55Q (VH: SEQ ID NO:113; VL: SEQ ID NO:168), несущие мутации Fc MLNS или MLNS + GAALIE. REGN10987 и REGN10933 были включены в качестве сравнения. Вкратце, антитела разбавляли в кинетическом буфере при 3 мкг/мл и загружали на сенсоры белка А в течение 75 секунд. После короткого этапа уравновешивания в кинетическом буфере загруженные сенсоры перемещали в лунки, содержащие варианты RBD, при 5 мкг/мл в кинетическом буфере и фиксировали ассоциацию в течение 3 минут. Диссоциацию комплекса проводили в кинетическом буфере в течение 3 минут. Данные показаны на фиг. 71A-71B; "WT" = Wuhan-Hu-1 с D614G; "Тройной мутант" в нижнем ряду = Wuhan-Hu-1 с D614G и добавлены южноафриканский вариант B.1.351 с мутациями RBD K417N, E484K и N501Y. Другие мутации, присутствующие в южнойафриканском варианте B.1.351, не присутствовали в тестируемом RBD «SA».
Нейтрализацию с помощью антител S309 против вариантов SARS-CoV-2 оценивали с использованием псевдотипированного вируса MLV и клеток-мишеней Vero-E6, экспрессирующих TMPRSS2. Оценивали S309 (VH: SEQ ID NO:105; VL: SEQ ID NO:168) с Fc дикого типа и S309 N55Q (VH: SEQ ID NO:113; VL: SEQ ID NO:168), несущие мутации Fc MLNS или MLNS + GAALIE. Также оценивали REGN10987, REGN10933 и комбинацию REGN10987 + REGN10933. Данные показаны на фиг. 72. "WT" означает Wuhan-Hu-1; "UK" означает SARS-CoV-2 вариант B.1.1.7; и "SA" означает вариант B.1.351.
Различные варианты осуществления, описанные в настоящем документе, можно объединять, получая дополнительные варианты осуществления. Все патенты США, публикации патентных заявок США, патентные заявки США, иностранные патенты, иностранные патентные заявки и непатентные публикации, указанные в настоящем описании и/или перечисленные информационном листе заявки, включая патентную заявку США 62/981,984, поданную 26 февраля, 2020, патентную заявку США 62/982,661, поданную 27 февраля, 2020, патентную заявку США 62/987,298, поданную 9 марта, 2020, патентную заявку США 62/989,522, поданную 13 марта, 2020, патентную заявку США 62/990,369, поданную 16 марта, 2020, патентную заявку США 62/992,082, поданную 19 марта, 2020, патентную заявку США 62/994,235, поданную 24 марта, 2020, патентную заявку США 63/001,204, поданную 27 марта, 2020, патентную заявку США 63/003,214, поданную 31 марта, 2020, патентную заявку США 63/005,206, поданную 3 апреля, 2020, патентную заявку США 63/010,589, поданную 15 апреля, 2020, патентную заявку США 63/011,971, поданную 17 апреля, 2020, патентную заявку США 63/014,024, поданную 22 апреля, 2020, патентную заявку США 63/023,788, поданную 12 мая, 2020, патентную заявку США 63/025,133, поданную 14 мая, 2020, патентную заявку США 63/039,813, поданную 16 июня, 2020, патентную заявку США 63/043,653, поданную 24 июня, 2020, патентную заявку США 63/050,331, поданную 10 июля, 2020, и патентную заявку США 63/052,810, поданную 16 июля, 2020, полностью включены в настоящий документ посредством ссылки. Аспекты вариантов осуществления может быть модифицировано, если необходимо использовать концепции различных патентов, заявок и публикаций для обеспечения еще нескольких дополниительных вариантов осуществления.
В настоящий вариант осуществления можно вносить указанные и другие изменения в свете вышеприведенного подробного описания. В целом, термины, используемые в следующей формуле изобретения, не должны толковаться как ограничивающие формулу изобретения конкретными вариантами осуществления, раскрытыми в описании и формуле изобретения, но должны толковаться как включающие все возможные варианты осуществления вместе с полным объемом эквивалентов, на которые распространяется такая формула изобретения. Соответственно, формула изобретения не ограничена описанием.
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> ВИР БАЙОТЕКНОЛОДЖИ, ИНК.
ХУМАБС БИОМЕД СА
<120> АНТИТЕЛА ПРОТИВ SARS-COV-2 И СПОСОБЫ ИХ ПРИМЕНЕНИЯ
<130> 930585.402WO
<140> PCT
<141> 2021-02-25
<150> US 62/981,984
<151> 2020-02-26
<150> US 62/982,661
<151> 2020-02-27
<150> US 62/987,298
<151> 2020-03-09
<150> US 62/989,522
<151> 2020-03-13
<150> US 62/990,369
<151> 2020-03-16
<150> US 62/992,082
<151> 2020-03-19
<150> US 62/994,235
<151> 2020-03-24
<150> US 63/001,204
<151> 2020-03-27
<150> US 63/003,214
<151> 2020-03-31
<150> US 63/005,206
<151> 2020-04-03
<150> US 63/010,589
<151> 2020-04-15
<150> US 63/011,971
<151> 2020-04-17
<150> US 63/014,024
<151> 2020-04-22
<150> US 63/023,788
<151> 2020-05-12
<150> US 63/025,133
<151> 2020-05-14
<150> US 63/039,813
<151> 2020-06-16
<150> US 63/043,653
<151> 2020-06-24
<150> US 63/050,331
<151> 2020-07-10
<150> US 63/052,810
<151> 2020-07-16
<160> 267
<170> FastSEQ для Windows версии 4.0
<210> 1
<211> 123
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S300-v1 mAb SARS-CoV-2
<400> 1
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Leu
35 40 45
Gly Trp Val Asn Gly Tyr Ser Gly Ala Thr Arg Tyr Ala Gln Lys Tyr
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Arg Leu Arg Pro Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Arg Pro Ser His Glu Trp Ala Met Tyr Phe Phe Asp Asn
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 2
<211> 8
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH1 S300-v1 mAb SARS-CoV-2
<400> 2
Gly Tyr Thr Phe Thr Asp Tyr Tyr
1 5
<210> 3
<211> 8
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH2 S300-v1 mAb SARS-CoV-2
<400> 3
Val Asn Gly Tyr Ser Gly Ala Thr
1 5
<210> 4
<211> 16
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH3 S300-v1 mAb SARS-CoV-2
<400> 4
Ala Arg Asp Arg Pro Ser His Glu Trp Ala Met Tyr Phe Phe Asp Asn
1 5 10 15
<210> 5
<211> 109
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VL (VK) S300-v1 mAb SARS-CoV-2
<400> 5
Gln Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Pro Ser Ser
20 25 30
Cys Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Gly Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Arg Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Ser Ser Pro
85 90 95
Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 6
<211> 7
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRL1 S300-v1 mAb SARS-CoV-2
<400> 6
Gln Ser Val Pro Ser Ser Cys
1 5
<210> 7
<211> 3
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRL2 S300-v1 mAb SARS-CoV-2
<400> 7
Gly Ala Ser
1
<210> 8
<211> 10
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRL3 S300-v1 mAb SARS-CoV-2
<400> 8
Gln Gln Tyr Gly Ser Ser Pro Pro Leu Thr
1 5 10
<210> 9
<211> 123
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S300-v1.1 mAb SARS-CoV-2
<400> 9
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Leu
35 40 45
Gly Trp Val Gln Gly Tyr Ser Gly Ala Thr Arg Tyr Ala Gln Lys Tyr
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Arg Leu Arg Pro Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Arg Pro Ser His Glu Trp Ala Met Tyr Phe Phe Asp Asn
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 10
<211> 123
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S300-v1.2 mAb SARS-CoV-2
<400> 10
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Leu
35 40 45
Gly Trp Val Asn Ala Tyr Ser Gly Ala Thr Arg Tyr Ala Gln Lys Tyr
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Arg Leu Arg Pro Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Arg Pro Ser His Glu Trp Ala Met Tyr Phe Phe Asp Asn
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 11
<211> 123
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S300-v1.3 mAb SARS-CoV-2
<400> 11
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Leu
35 40 45
Gly Trp Val Asn Ser Tyr Ser Gly Ala Thr Arg Tyr Ala Gln Lys Tyr
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Arg Leu Arg Pro Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Arg Pro Ser His Glu Trp Ala Met Tyr Phe Phe Asp Asn
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 12
<211> 123
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S300-v1.4 mAb SARS-CoV-2
<400> 12
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Leu
35 40 45
Gly Trp Val Asn Pro Tyr Ser Gly Ala Thr Arg Tyr Ala Gln Lys Tyr
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Arg Leu Arg Pro Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Arg Pro Ser His Glu Trp Ala Met Tyr Phe Phe Asp Asn
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 13
<211> 123
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S300-v1.5 mAb SARS-CoV-2
<400> 13
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Leu
35 40 45
Gly Trp Val Asn Gln Tyr Ser Gly Ala Thr Arg Tyr Ala Gln Lys Tyr
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Arg Leu Arg Pro Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Arg Pro Ser His Glu Trp Ala Met Tyr Phe Phe Asp Asn
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 14
<211> 123
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S300-v1.6 mAb SARS-CoV-2
<400> 14
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Leu
35 40 45
Gly Trp Val Leu Gly Tyr Ser Gly Ala Thr Arg Tyr Ala Gln Lys Tyr
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Arg Leu Arg Pro Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Arg Pro Ser His Glu Trp Ala Met Tyr Phe Phe Asp Asn
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 15
<211> 123
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S300-v1.7 mAb SARS-CoV-2
<400> 15
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Leu
35 40 45
Gly Trp Val Thr Gly Tyr Ser Gly Ala Thr Arg Tyr Ala Gln Lys Tyr
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Arg Leu Arg Pro Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Arg Pro Ser His Glu Trp Ala Met Tyr Phe Phe Asp Asn
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 16
<211> 8
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH2 S300-v1.1 mAb SARS-CoV-2
<400> 16
Val Gln Gly Tyr Ser Gly Ala Thr
1 5
<210> 17
<211> 8
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH2 S300-v1.2 mAb SARS-CoV-2
<400> 17
Val Asn Ala Tyr Ser Gly Ala Thr
1 5
<210> 18
<211> 8
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH2 S300-v1.3 mAb SARS-CoV-2
<400> 18
Val Asn Ser Tyr Ser Gly Ala Thr
1 5
<210> 19
<211> 8
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH2 S300-v1.4 mAb SARS-CoV-2
<400> 19
Val Asn Pro Tyr Ser Gly Ala Thr
1 5
<210> 20
<211> 8
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH2 S300-v1.5 mAb SARS-CoV-2
<400> 20
Val Asn Gln Tyr Ser Gly Ala Thr
1 5
<210> 21
<211> 8
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH2 S300-v1.6 mAb SARS-CoV-2
<400> 21
Val Leu Gly Tyr Ser Gly Ala Thr
1 5
<210> 22
<211> 8
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH2 S300-v1.7 mAb SARS-CoV-2
<400> 22
Val Thr Gly Tyr Ser Gly Ala Thr
1 5
<210> 23
<211> 123
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S300-v1.8 mAb SARS-CoV-2
<400> 23
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Leu
35 40 45
Gly Trp Val Asn Gly Tyr Ser Gly Ala Thr Arg Tyr Ala Gln Lys Tyr
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Arg Leu Arg Pro Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Arg Pro Ser His Glu Phe Ala Met Tyr Phe Phe Asp Asn
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 24
<211> 123
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S300-v1.9 mAb SARS-CoV-2
<400> 24
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Leu
35 40 45
Gly Trp Val Asn Gly Tyr Ser Gly Ala Thr Arg Tyr Ala Gln Lys Tyr
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Arg Leu Arg Pro Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Arg Pro Ser His Glu Tyr Ala Met Tyr Phe Phe Asp Asn
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 25
<211> 16
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH3 S300-v1.8 mAb SARS-CoV-2
<400> 25
Ala Arg Asp Arg Pro Ser His Glu Phe Ala Met Tyr Phe Phe Asp Asn
1 5 10 15
<210> 26
<211> 16
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH3 S300-v1.9 mAb SARS-CoV-2
<400> 26
Ala Arg Asp Arg Pro Ser His Glu Tyr Ala Met Tyr Phe Phe Asp Asn
1 5 10 15
<210> 27
<211> 123
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S300-v2 mAb SARS-CoV-2
<400> 27
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Leu
35 40 45
Gly Phe Val Asn Gly Tyr Ser Gly Ala Thr Arg Tyr Ala Gln Lys Tyr
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Arg Leu Arg Pro Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Arg Pro Ser His Glu Trp Ala Met Tyr Phe Phe Asp Asn
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 28
<211> 123
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S300-v2.1 mAb SARS-CoV-2
<400> 28
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Leu
35 40 45
Gly Phe Val Gln Gly Tyr Ser Gly Ala Thr Arg Tyr Ala Gln Lys Tyr
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Arg Leu Arg Pro Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Arg Pro Ser His Glu Trp Ala Met Tyr Phe Phe Asp Asn
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 29
<211> 123
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S300-v2.2 mAb SARS-CoV-2
<400> 29
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Leu
35 40 45
Gly Phe Val Asn Ala Tyr Ser Gly Ala Thr Arg Tyr Ala Gln Lys Tyr
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Arg Leu Arg Pro Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Arg Pro Ser His Glu Trp Ala Met Tyr Phe Phe Asp Asn
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 30
<211> 123
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S300-v2.3 mAb SARS-CoV-2
<400> 30
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Leu
35 40 45
Gly Phe Val Asn Ser Tyr Ser Gly Ala Thr Arg Tyr Ala Gln Lys Tyr
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Arg Leu Arg Pro Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Arg Pro Ser His Glu Trp Ala Met Tyr Phe Phe Asp Asn
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 31
<211> 123
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S300-v2.4 mAb SARS-CoV-2
<400> 31
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Leu
35 40 45
Gly Phe Val Asn Pro Tyr Ser Gly Ala Thr Arg Tyr Ala Gln Lys Tyr
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Arg Leu Arg Pro Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Arg Pro Ser His Glu Trp Ala Met Tyr Phe Phe Asp Asn
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 32
<211> 123
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S300-v2.5 mAb SARS-CoV-2
<400> 32
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Leu
35 40 45
Gly Phe Val Asn Gln Tyr Ser Gly Ala Thr Arg Tyr Ala Gln Lys Tyr
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Arg Leu Arg Pro Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Arg Pro Ser His Glu Trp Ala Met Tyr Phe Phe Asp Asn
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 33
<211> 123
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S300-v2.6 mAb SARS-CoV-2
<400> 33
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Leu
35 40 45
Gly Phe Val Leu Gly Tyr Ser Gly Ala Thr Arg Tyr Ala Gln Lys Tyr
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Arg Leu Arg Pro Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Arg Pro Ser His Glu Trp Ala Met Tyr Phe Phe Asp Asn
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 34
<211> 123
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S300-v2.7 mAb SARS-CoV-2
<400> 34
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Leu
35 40 45
Gly Phe Val Thr Gly Tyr Ser Gly Ala Thr Arg Tyr Ala Gln Lys Tyr
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Arg Leu Arg Pro Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Arg Pro Ser His Glu Trp Ala Met Tyr Phe Phe Asp Asn
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 35
<211> 123
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S300-v2.8 mAb SARS-CoV-2
<400> 35
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Leu
35 40 45
Gly Phe Val Asn Gly Tyr Ser Gly Ala Thr Arg Tyr Ala Gln Lys Tyr
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Arg Leu Arg Pro Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Arg Pro Ser His Glu Phe Ala Met Tyr Phe Phe Asp Asn
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 36
<211> 123
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S300-v2.9 mAb SARS-CoV-2
<400> 36
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Leu
35 40 45
Gly Phe Val Asn Gly Tyr Ser Gly Ala Thr Arg Tyr Ala Gln Lys Tyr
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Arg Leu Arg Pro Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Arg Pro Ser His Glu Tyr Ala Met Tyr Phe Phe Asp Asn
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 37
<211> 123
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S300-v3 mAb SARS-CoV-2
<400> 37
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Leu
35 40 45
Gly Tyr Val Asn Gly Tyr Ser Gly Ala Thr Arg Tyr Ala Gln Lys Tyr
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Arg Leu Arg Pro Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Arg Pro Ser His Glu Trp Ala Met Tyr Phe Phe Asp Asn
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 38
<211> 123
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S300-v3.1 mAb SARS-CoV-2
<400> 38
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Leu
35 40 45
Gly Tyr Val Gln Gly Tyr Ser Gly Ala Thr Arg Tyr Ala Gln Lys Tyr
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Arg Leu Arg Pro Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Arg Pro Ser His Glu Trp Ala Met Tyr Phe Phe Asp Asn
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 39
<211> 123
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S300-v3.2 mAb SARS-CoV-2
<400> 39
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Leu
35 40 45
Gly Tyr Val Asn Ala Tyr Ser Gly Ala Thr Arg Tyr Ala Gln Lys Tyr
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Arg Leu Arg Pro Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Arg Pro Ser His Glu Trp Ala Met Tyr Phe Phe Asp Asn
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 40
<211> 123
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S300-v3.3 mAb SARS-CoV-2
<400> 40
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Leu
35 40 45
Gly Tyr Val Asn Ser Tyr Ser Gly Ala Thr Arg Tyr Ala Gln Lys Tyr
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Arg Leu Arg Pro Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Arg Pro Ser His Glu Trp Ala Met Tyr Phe Phe Asp Asn
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 41
<211> 123
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S300-v3.4 mAb SARS-CoV-2
<400> 41
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Leu
35 40 45
Gly Tyr Val Asn Pro Tyr Ser Gly Ala Thr Arg Tyr Ala Gln Lys Tyr
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Arg Leu Arg Pro Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Arg Pro Ser His Glu Trp Ala Met Tyr Phe Phe Asp Asn
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 42
<211> 123
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S300-v3.5 mAb SARS-CoV-2
<400> 42
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Leu
35 40 45
Gly Tyr Val Asn Gln Tyr Ser Gly Ala Thr Arg Tyr Ala Gln Lys Tyr
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Arg Leu Arg Pro Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Arg Pro Ser His Glu Trp Ala Met Tyr Phe Phe Asp Asn
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 43
<211> 123
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S300-v3.6 mAb SARS-CoV-2
<400> 43
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Leu
35 40 45
Gly Tyr Val Leu Gly Tyr Ser Gly Ala Thr Arg Tyr Ala Gln Lys Tyr
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Arg Leu Arg Pro Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Arg Pro Ser His Glu Trp Ala Met Tyr Phe Phe Asp Asn
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 44
<211> 123
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S300-v3.7 mAb SARS-CoV-2
<400> 44
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Leu
35 40 45
Gly Tyr Val Thr Gly Tyr Ser Gly Ala Thr Arg Tyr Ala Gln Lys Tyr
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Arg Leu Arg Pro Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Arg Pro Ser His Glu Trp Ala Met Tyr Phe Phe Asp Asn
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 45
<211> 123
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S300-v3.8 mAb SARS-CoV-2
<400> 45
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Leu
35 40 45
Gly Tyr Val Asn Gly Tyr Ser Gly Ala Thr Arg Tyr Ala Gln Lys Tyr
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Arg Leu Arg Pro Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Arg Pro Ser His Glu Phe Ala Met Tyr Phe Phe Asp Asn
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 46
<211> 123
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S300-v3.9 mAb SARS-CoV-2
<400> 46
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Leu
35 40 45
Gly Tyr Val Asn Gly Tyr Ser Gly Ala Thr Arg Tyr Ala Gln Lys Tyr
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Arg Leu Arg Pro Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Arg Pro Ser His Glu Tyr Ala Met Tyr Phe Phe Asp Asn
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 47
<211> 109
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VL (VK) S300-v10 mAb SARS-CoV-2
<400> 47
Gln Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Pro Ser Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Gly Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Arg Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Ser Ser Pro
85 90 95
Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 48
<211> 109
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VL (VK) S300-v11 mAb SARS-CoV-2
<400> 48
Gln Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Pro Ser Ser
20 25 30
Ser Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Gly Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Arg Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Ser Ser Pro
85 90 95
Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 49
<211> 109
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VL (VK) S300-v12 mAb SARS-CoV-2
<400> 49
Gln Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Pro Ser Ser
20 25 30
Thr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Gly Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Arg Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Ser Ser Pro
85 90 95
Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 50
<211> 109
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VL (VK) S300-v13 mAb SARS-CoV-2
<400> 50
Gln Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Pro Ser Ser
20 25 30
Ala Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Gly Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Arg Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Ser Ser Pro
85 90 95
Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 51
<211> 7
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRL1 S300-v10 mAb SARS-CoV-2
<400> 51
Gln Ser Val Pro Ser Ser Tyr
1 5
<210> 52
<211> 7
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRL1 S300-v11 mAb SARS-CoV-2
<400> 52
Gln Ser Val Pro Ser Ser Ser
1 5
<210> 53
<211> 7
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRL1 S300-v12 mAb SARS-CoV-2
<400> 53
Gln Ser Val Pro Ser Ser Thr
1 5
<210> 54
<211> 7
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRL1 S300-v13 mAb SARS-CoV-2
<400> 54
Gln Ser Val Pro Ser Ser Ala
1 5
<210> 55
<211> 121
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S302 mAb SARS-CoV-2
<400> 55
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Ser Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Asp Ile Ser Ser Gly Trp Asp Arg Val Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 56
<211> 8
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH1 S302 mAb SARS-CoV-2
<400> 56
Gly Phe Thr Phe Ser Ser Tyr Gly
1 5
<210> 57
<211> 8
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH2 S302 mAb SARS-CoV-2
<400> 57
Ile Ser Tyr Asp Gly Ser Asn Lys
1 5
<210> 58
<211> 14
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH3 S302 mAb SARS-CoV-2
<400> 58
Ala Lys Asp Ile Ser Ser Gly Trp Asp Arg Val Phe Asp Tyr
1 5 10
<210> 59
<211> 108
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VL (VK) S302 mAb SARS-CoV-2
<400> 59
Glu Ile Leu Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Thr Ser Gln Ser Val Gly Ser Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Ala Ala Ser Ser Arg Ala Ile Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Ser Ser Pro
85 90 95
Trp Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 60
<211> 7
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRL1 S302 mAb SARS-CoV-2
<400> 60
Gln Ser Val Gly Ser Ser Tyr
1 5
<210> 61
<211> 3
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRL2 S302 mAb SARS-CoV-2
<400> 61
Ala Ala Ser
1
<210> 62
<211> 9
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRL3 S302 mAb SARS-CoV-2
<400> 62
Gln Gln Tyr Gly Ser Ser Pro Trp Thr
1 5
<210> 63
<211> 124
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S303-v1 mAb SARS-CoV-2
<400> 63
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Leu Thr Tyr
20 25 30
Ser Met Asn Trp Val Arg Gln Thr Pro Gly Lys Arg Leu Gln Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ala Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Thr Val Thr Ala Asp Asp Thr Ala Ile Tyr Phe Cys
85 90 95
Ala Arg Glu Arg Asp Asp Ile Phe Pro Met Gly Leu Asn Ala Phe Asp
100 105 110
Ile Trp Gly Gln Gly Ala Met Val Ile Val Ser Ser
115 120
<210> 64
<211> 8
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH1 S303-v1 mAb SARS-CoV-2
<400> 64
Gly Phe Thr Phe Leu Thr Tyr Ser
1 5
<210> 65
<211> 8
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH2 S303-v1 mAb SARS-CoV-2
<400> 65
Ile Ser Gly Ser Gly Gly Ala Thr
1 5
<210> 66
<211> 17
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH3 S303-v1 mAb SARS-CoV-2
<400> 66
Ala Arg Glu Arg Asp Asp Ile Phe Pro Met Gly Leu Asn Ala Phe Asp
1 5 10 15
Ile
<210> 67
<211> 107
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VL (VK) S303-v1 mAb SARS-CoV-2
<400> 67
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Asn Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Ser Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Ser Ala Thr Tyr Tyr Cys Gln Gln Tyr Asp Thr Tyr Ser Trp
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 68
<211> 6
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRL1 S303-v1 mAb SARS-CoV-2
<400> 68
Gln Ser Ile Ser Asn Trp
1 5
<210> 69
<211> 3
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRL2 S303-v1 mAb SARS-CoV-2
<400> 69
Lys Ala Ser
1
<210> 70
<211> 9
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRL3 S303-v1 mAb SARS-CoV-2
<400> 70
Gln Gln Tyr Asp Thr Tyr Ser Trp Thr
1 5
<210> 71
<211> 107
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VL (VK) S303-v2 mAb SARS-CoV-2
<400> 71
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Asn Phe
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Ser Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Ser Ala Thr Tyr Tyr Cys Gln Gln Tyr Asp Thr Tyr Ser Trp
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 72
<211> 107
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VL (VK) S303-v3 mAb SARS-CoV-2
<400> 72
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Asn Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Ser Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Ser Ala Thr Tyr Tyr Cys Gln Gln Tyr Asp Thr Tyr Ser Trp
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 73
<211> 6
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRL1 S303-v2 mAb SARS-CoV-2
<400> 73
Gln Ser Ile Ser Asn Phe
1 5
<210> 74
<211> 6
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRL1 S303-v3 mAb SARS-CoV-2
<400> 74
Gln Ser Ile Ser Asn Tyr
1 5
<210> 75
<211> 107
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VL (VK) S303-v4 mAb SARS-CoV-2
<400> 75
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Asn Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Ser Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Ser Ala Thr Tyr Tyr Cys Gln Gln Tyr Asp Thr Tyr Ser Phe
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 76
<211> 107
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VL (VK) S303-v5 mAb SARS-CoV-2
<400> 76
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Asn Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Ser Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Ser Ala Thr Tyr Tyr Cys Gln Gln Tyr Asp Thr Tyr Ser Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 77
<211> 9
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRL3 S303-v4 mAb SARS-CoV-2
<400> 77
Gln Gln Tyr Asp Thr Tyr Ser Phe Thr
1 5
<210> 78
<211> 9
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRL3 S303-v5 mAb SARS-CoV-2
<400> 78
Gln Gln Tyr Asp Thr Tyr Ser Tyr Thr
1 5
<210> 79
<211> 120
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S304 mAb SARS-CoV-2
<400> 79
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Asp Met His Trp Val Arg Gln Thr Thr Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Thr Ile Gly Thr Ala Gly Asp Thr Tyr Tyr Pro Asp Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Glu Asp Ala Lys Asn Ser Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Gly Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Gly Asp Ser Ser Gly Tyr Tyr Tyr Tyr Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Leu Thr Val Ser Ser
115 120
<210> 80
<211> 8
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH1 S304 mAb SARS-CoV-2
<400> 80
Gly Phe Thr Phe Ser Ser Tyr Asp
1 5
<210> 81
<211> 7
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH2 S304 mAb SARS-CoV-2
<400> 81
Ile Gly Thr Ala Gly Asp Thr
1 5
<210> 82
<211> 14
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH3 S304 mAb SARS-CoV-2
<400> 82
Ala Arg Gly Asp Ser Ser Gly Tyr Tyr Tyr Tyr Phe Asp Tyr
1 5 10
<210> 83
<211> 108
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VL (VK) S304 mAb SARS-CoV-2
<400> 83
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ala Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Gly Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Ile Tyr Tyr Cys Gln Gln Ser Tyr Val Ser Pro Thr
85 90 95
Tyr Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys
100 105
<210> 84
<211> 6
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRL1 S304 mAb SARS-CoV-2
<400> 84
Gln Ser Ile Gly Ser Tyr
1 5
<210> 85
<211> 3
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRL2 S304 mAb SARS-CoV-2
<400> 85
Ala Ala Ser
1
<210> 86
<211> 10
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRL3 S304 mAb SARS-CoV-2
<400> 86
Gln Gln Ser Tyr Val Ser Pro Thr Tyr Thr
1 5 10
<210> 87
<211> 123
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S306 mAb SARS-CoV-2
<400> 87
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Thr Tyr Thr Phe Thr Ser Phe
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Thr Thr Tyr Ser Gly Asp Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Asn Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ser Asp Tyr Phe Asp Ser Ser Gly Tyr Tyr His Ser Phe Asp Tyr
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 88
<211> 8
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH1 S306 mAb SARS-CoV-2
<400> 88
Thr Tyr Thr Phe Thr Ser Phe Gly
1 5
<210> 89
<211> 8
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH2 S306 mAb SARS-CoV-2
<400> 89
Ile Thr Thr Tyr Ser Gly Asp Thr
1 5
<210> 90
<211> 16
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH3 S306 mAb SARS-CoV-2
<400> 90
Ala Ser Asp Tyr Phe Asp Ser Ser Gly Tyr Tyr His Ser Phe Asp Tyr
1 5 10 15
<210> 91
<211> 109
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VL (VK) S306 mAb SARS-CoV-2
<400> 91
Glu Ile Val Leu Thr Gln Ser Pro Asp Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Arg Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Lys Arg Ala Thr Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Arg Ser Asn Trp Pro Pro
85 90 95
Gly Cys Ser Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 92
<211> 6
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRL1 S306 mAb SARS-CoV-2
<400> 92
Gln Ser Val Ser Ser Tyr
1 5
<210> 93
<211> 3
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRL2 S306 mAb SARS-CoV-2
<400> 93
Asp Ala Ser
1
<210> 94
<211> 11
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRL3 S306 mAb SARS-CoV-2
<400> 94
Gln Gln Arg Ser Asn Trp Pro Pro Gly Cys Ser
1 5 10
<210> 95
<211> 127
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S308-v1 mAb SARS-CoV-2
<400> 95
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Arg Phe Thr Phe Ser Ser Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Trp His Asp Gly Asn Asn Lys His Tyr Gly Asp Ser Val
50 55 60
Lys Gly Arg Val Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Thr Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ala Val Thr Thr Phe Lys Gly Ser Gly Arg Ala Arg Met Arg
100 105 110
Gly Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120 125
<210> 96
<211> 8
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH1 S308-v1 mAb SARS-CoV-2
<400> 96
Arg Phe Thr Phe Ser Ser Tyr Gly
1 5
<210> 97
<211> 8
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH2 S308-v1 mAb SARS-CoV-2
<400> 97
Ile Trp His Asp Gly Asn Asn Lys
1 5
<210> 98
<211> 20
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH3 S308-v1 mAb SARS-CoV-2
<400> 98
Ala Arg Ala Val Thr Thr Phe Lys Gly Ser Gly Arg Ala Arg Met Arg
1 5 10 15
Gly Met Asp Val
20
<210> 99
<211> 107
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VL (VK) S308-v1 mAb SARS-CoV-2
<400> 99
Asp Ile Gln Leu Thr Gln Ser Pro Ser Phe Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Asn Thr Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln His Leu Asp Thr Tyr Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys
100 105
<210> 100
<211> 6
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRL1 S308-v1 mAb SARS-CoV-2
<400> 100
Gln Gly Ile Asn Thr Tyr
1 5
<210> 101
<211> 3
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRL2 S308-v1 mAb SARS-CoV-2
<400> 101
Ala Ala Ser
1
<210> 102
<211> 9
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRL3 S308-v1 mAb SARS-CoV-2
<400> 102
Gln His Leu Asp Thr Tyr Pro Phe Thr
1 5
<210> 103
<211> 127
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S308-v2 mAb SARS-CoV-2
<400> 103
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Arg Phe Thr Phe Ser Ser Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Trp His Asp Gly Asn Asn Lys His Tyr Gly Asp Ser Val
50 55 60
Lys Gly Arg Val Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Thr Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ala Val Thr Thr Phe Lys Gly Ser Gly Arg Ala Arg Leu Arg
100 105 110
Gly Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120 125
<210> 104
<211> 20
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH3 S308-v2 mAb SARS-CoV-2
<400> 104
Ala Arg Ala Val Thr Thr Phe Lys Gly Ser Gly Arg Ala Arg Leu Arg
1 5 10 15
Gly Met Asp Val
20
<210> 105
<211> 127
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S309-v1 mAb SARS-CoV-2
<400> 105
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Thr Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr
65 70 75 80
Met Glu Leu Arg Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Thr Arg Gly Ala Trp Phe Gly Glu Ser Leu Ile Gly
100 105 110
Gly Phe Asp Asn Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 106
<211> 8
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH1 S309-v1 mAb SARS-CoV-2
<400> 106
Gly Tyr Pro Phe Thr Ser Tyr Gly
1 5
<210> 107
<211> 8
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH2 S309-v1 mAb SARS-CoV-2
<400> 107
Ile Ser Thr Tyr Asn Gly Asn Thr
1 5
<210> 108
<211> 20
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH3 S309-v1 mAb SARS-CoV-2
<400> 108
Ala Arg Asp Tyr Thr Arg Gly Ala Trp Phe Gly Glu Ser Leu Ile Gly
1 5 10 15
Gly Phe Asp Asn
20
<210> 109
<211> 107
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VL (VK) S309-v1 mAb SARS-CoV-2
<400> 109
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Thr Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Asn Asn Tyr
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Lys Leu Leu Ile
35 40 45
Tyr Gly Ala Ser Thr Leu Gln Ser Gly Val Pro Ser Arg Phe Arg Gly
50 55 60
Ser Gly Ser Gly Thr Gly Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Val Ala Ser Tyr Tyr Cys Arg Lys Tyr Asn Ser Ala Pro Trp
85 90 95
Thr Phe Gly Gln Gly Thr Arg Val Glu Ile Lys
100 105
<210> 110
<211> 6
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRL1 S309-v1 mAb SARS-CoV-2
<400> 110
Gln Gly Ile Asn Asn Tyr
1 5
<210> 111
<211> 3
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRL2 S309-v1 mAb SARS-CoV-2
<400> 111
Gly Ala Ser
1
<210> 112
<211> 9
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRL3 S309-v1 mAb SARS-CoV-2
<400> 112
Arg Lys Tyr Asn Ser Ala Pro Trp Thr
1 5
<210> 113
<211> 127
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S309-v1.1 mAb SARS-CoV-2
<400> 113
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Thr Tyr Gln Gly Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr
65 70 75 80
Met Glu Leu Arg Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Thr Arg Gly Ala Trp Phe Gly Glu Ser Leu Ile Gly
100 105 110
Gly Phe Asp Asn Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 114
<211> 127
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S309-v1.2 mAb SARS-CoV-2
<400> 114
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Thr Tyr Asn Ser Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr
65 70 75 80
Met Glu Leu Arg Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Thr Arg Gly Ala Trp Phe Gly Glu Ser Leu Ile Gly
100 105 110
Gly Phe Asp Asn Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 115
<211> 127
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S309-v1.3 mAb SARS-CoV-2
<400> 115
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Thr Tyr Asn Ala Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr
65 70 75 80
Met Glu Leu Arg Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Thr Arg Gly Ala Trp Phe Gly Glu Ser Leu Ile Gly
100 105 110
Gly Phe Asp Asn Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 116
<211> 127
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S309-v1.4 mAb SARS-CoV-2
<400> 116
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Thr Tyr Asn Gln Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr
65 70 75 80
Met Glu Leu Arg Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Thr Arg Gly Ala Trp Phe Gly Glu Ser Leu Ile Gly
100 105 110
Gly Phe Asp Asn Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 117
<211> 127
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S309-v1.5 mAb SARS-CoV-2
<400> 117
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Thr Tyr Leu Gly Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr
65 70 75 80
Met Glu Leu Arg Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Thr Arg Gly Ala Trp Phe Gly Glu Ser Leu Ile Gly
100 105 110
Gly Phe Asp Asn Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 118
<211> 127
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S309-v1.6 mAb SARS-CoV-2
<400> 118
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Thr Tyr Thr Gly Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr
65 70 75 80
Met Glu Leu Arg Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Thr Arg Gly Ala Trp Phe Gly Glu Ser Leu Ile Gly
100 105 110
Gly Phe Asp Asn Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 119
<211> 127
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S309-v1.7 mAb SARS-CoV-2
<400> 119
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Thr Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr
65 70 75 80
Met Glu Leu Arg Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Thr Arg Gly Ala Phe Phe Gly Glu Ser Leu Ile Gly
100 105 110
Gly Phe Asp Asn Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 120
<211> 127
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S309-v1.8 mAb SARS-CoV-2
<400> 120
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Thr Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr
65 70 75 80
Met Glu Leu Arg Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Thr Arg Gly Ala Tyr Phe Gly Glu Ser Leu Ile Gly
100 105 110
Gly Phe Asp Asn Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 121
<211> 8
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH2 S309-v1.1 mAb SARS-CoV-2
<400> 121
Ile Ser Thr Tyr Gln Gly Asn Thr
1 5
<210> 122
<211> 8
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH2 S309-v1.2 mAb SARS-CoV-2
<400> 122
Ile Ser Thr Tyr Asn Ser Asn Thr
1 5
<210> 123
<211> 8
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH2 S309-v1.3 mAb SARS-CoV-2
<400> 123
Ile Ser Thr Tyr Asn Ala Asn Thr
1 5
<210> 124
<211> 8
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH2 S309-v1.4 mAb SARS-CoV-2
<400> 124
Ile Ser Thr Tyr Asn Gln Asn Thr
1 5
<210> 125
<211> 8
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH2 S309-v1.5 mAb SARS-CoV-2
<400> 125
Ile Ser Thr Tyr Leu Gly Asn Thr
1 5
<210> 126
<211> 8
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH2 S309-v1.6 mAb SARS-CoV-2
<400> 126
Ile Ser Thr Tyr Thr Gly Asn Thr
1 5
<210> 127
<211> 20
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH3 S309-v1.7 mAb SARS-CoV-2
<400> 127
Ala Arg Asp Tyr Thr Arg Gly Ala Phe Phe Gly Glu Ser Leu Ile Gly
1 5 10 15
Gly Phe Asp Asn
20
<210> 128
<211> 20
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH3 S309-v1.8 mAb SARS-CoV-2
<400> 128
Ala Arg Asp Tyr Thr Arg Gly Ala Tyr Phe Gly Glu Ser Leu Ile Gly
1 5 10 15
Gly Phe Asp Asn
20
<210> 129
<211> 127
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S309-v2 mAb SARS-CoV-2
<400> 129
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Phe Ile Ser Thr Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr
65 70 75 80
Met Glu Leu Arg Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Thr Arg Gly Ala Trp Phe Gly Glu Ser Leu Ile Gly
100 105 110
Gly Phe Asp Asn Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 130
<211> 127
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S309-v2.1 mAb SARS-CoV-2
<400> 130
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Phe Ile Ser Thr Tyr Gln Gly Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr
65 70 75 80
Met Glu Leu Arg Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Thr Arg Gly Ala Trp Phe Gly Glu Ser Leu Ile Gly
100 105 110
Gly Phe Asp Asn Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 131
<211> 127
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S309-v2.2 mAb SARS-CoV-2
<400> 131
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Phe Ile Ser Thr Tyr Asn Ser Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr
65 70 75 80
Met Glu Leu Arg Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Thr Arg Gly Ala Trp Phe Gly Glu Ser Leu Ile Gly
100 105 110
Gly Phe Asp Asn Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 132
<211> 127
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S309-v2.3 mAb SARS-CoV-2
<400> 132
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Phe Ile Ser Thr Tyr Asn Ala Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr
65 70 75 80
Met Glu Leu Arg Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Thr Arg Gly Ala Trp Phe Gly Glu Ser Leu Ile Gly
100 105 110
Gly Phe Asp Asn Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 133
<211> 127
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S309-v2.4 mAb SARS-CoV-2
<400> 133
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Phe Ile Ser Thr Tyr Asn Gln Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr
65 70 75 80
Met Glu Leu Arg Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Thr Arg Gly Ala Trp Phe Gly Glu Ser Leu Ile Gly
100 105 110
Gly Phe Asp Asn Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 134
<211> 127
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S309-v2.5 mAb SARS-CoV-2
<400> 134
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Phe Ile Ser Thr Tyr Leu Gly Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr
65 70 75 80
Met Glu Leu Arg Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Thr Arg Gly Ala Trp Phe Gly Glu Ser Leu Ile Gly
100 105 110
Gly Phe Asp Asn Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 135
<211> 127
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S309-v2.6 mAb SARS-CoV-2
<400> 135
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Phe Ile Ser Thr Tyr Thr Gly Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr
65 70 75 80
Met Glu Leu Arg Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Thr Arg Gly Ala Trp Phe Gly Glu Ser Leu Ile Gly
100 105 110
Gly Phe Asp Asn Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 136
<211> 127
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S309-v2.7 mAb SARS-CoV-2
<400> 136
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Phe Ile Ser Thr Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr
65 70 75 80
Met Glu Leu Arg Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Thr Arg Gly Ala Phe Phe Gly Glu Ser Leu Ile Gly
100 105 110
Gly Phe Asp Asn Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 137
<211> 127
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S309-v2.8 mAb SARS-CoV-2
<400> 137
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Phe Ile Ser Thr Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr
65 70 75 80
Met Glu Leu Arg Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Thr Arg Gly Ala Tyr Phe Gly Glu Ser Leu Ile Gly
100 105 110
Gly Phe Asp Asn Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 138
<211> 127
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S309-v3 mAb SARS-CoV-2
<400> 138
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Tyr Ile Ser Thr Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr
65 70 75 80
Met Glu Leu Arg Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Thr Arg Gly Ala Trp Phe Gly Glu Ser Leu Ile Gly
100 105 110
Gly Phe Asp Asn Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 139
<211> 127
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S309-v3.1 mAb SARS-CoV-2
<400> 139
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Tyr Ile Ser Thr Tyr Gln Gly Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr
65 70 75 80
Met Glu Leu Arg Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Thr Arg Gly Ala Trp Phe Gly Glu Ser Leu Ile Gly
100 105 110
Gly Phe Asp Asn Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 140
<211> 127
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S309-v3.2 mAb SARS-CoV-2
<400> 140
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Tyr Ile Ser Thr Tyr Asn Ser Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr
65 70 75 80
Met Glu Leu Arg Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Thr Arg Gly Ala Trp Phe Gly Glu Ser Leu Ile Gly
100 105 110
Gly Phe Asp Asn Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 141
<211> 127
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S309-v3.3 mAb SARS-CoV-2
<400> 141
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Tyr Ile Ser Thr Tyr Asn Ala Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr
65 70 75 80
Met Glu Leu Arg Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Thr Arg Gly Ala Trp Phe Gly Glu Ser Leu Ile Gly
100 105 110
Gly Phe Asp Asn Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 142
<211> 127
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S309-v3.4 mAb SARS-CoV-2
<400> 142
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Tyr Ile Ser Thr Tyr Asn Gln Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr
65 70 75 80
Met Glu Leu Arg Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Thr Arg Gly Ala Trp Phe Gly Glu Ser Leu Ile Gly
100 105 110
Gly Phe Asp Asn Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 143
<211> 127
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S309-v3.5 mAb SARS-CoV-2
<400> 143
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Tyr Ile Ser Thr Tyr Leu Gly Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr
65 70 75 80
Met Glu Leu Arg Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Thr Arg Gly Ala Trp Phe Gly Glu Ser Leu Ile Gly
100 105 110
Gly Phe Asp Asn Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 144
<211> 127
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S309-v3.6 mAb SARS-CoV-2
<400> 144
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Tyr Ile Ser Thr Tyr Thr Gly Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr
65 70 75 80
Met Glu Leu Arg Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Thr Arg Gly Ala Trp Phe Gly Glu Ser Leu Ile Gly
100 105 110
Gly Phe Asp Asn Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 145
<211> 127
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S309-v3.7 mAb SARS-CoV-2
<400> 145
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Tyr Ile Ser Thr Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr
65 70 75 80
Met Glu Leu Arg Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Thr Arg Gly Ala Phe Phe Gly Glu Ser Leu Ile Gly
100 105 110
Gly Phe Asp Asn Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 146
<211> 127
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S309-v3.8 mAb SARS-CoV-2
<400> 146
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Tyr Ile Ser Thr Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr
65 70 75 80
Met Glu Leu Arg Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Thr Arg Gly Ala Tyr Phe Gly Glu Ser Leu Ile Gly
100 105 110
Gly Phe Asp Asn Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 147
<211> 107
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VL (VK) S309-v9 mAb SARS-CoV-2
<400> 147
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Thr Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Asn Asn Tyr
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Lys Leu Leu Ile
35 40 45
Tyr Gly Ala Ser Thr Leu Gln Ser Gly Val Pro Ser Arg Phe Arg Gly
50 55 60
Ser Gly Ser Gly Thr Gly Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Val Ala Ser Tyr Tyr Cys Arg Lys Tyr Asn Ser Ala Pro Gly
85 90 95
Thr Phe Gly Gln Gly Thr Arg Val Glu Ile Lys
100 105
<210> 148
<211> 107
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VL (VK) S309-v10 mAb SARS-CoV-2
<400> 148
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Thr Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Asn Asn Tyr
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Lys Leu Leu Ile
35 40 45
Tyr Gly Ala Ser Thr Leu Gln Ser Gly Val Pro Ser Arg Phe Arg Gly
50 55 60
Ser Gly Ser Gly Thr Gly Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Val Ala Ser Tyr Tyr Cys Arg Lys Tyr Asn Ser Ala Pro Arg
85 90 95
Thr Phe Gly Gln Gly Thr Arg Val Glu Ile Lys
100 105
<210> 149
<211> 107
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VL (VK) S309-v11 mAb SARS-CoV-2
<400> 149
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Thr Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Asn Asn Tyr
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Lys Leu Leu Ile
35 40 45
Tyr Gly Ala Ser Thr Leu Gln Ser Gly Val Pro Ser Arg Phe Arg Gly
50 55 60
Ser Gly Ser Gly Thr Gly Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Val Ala Ser Tyr Tyr Cys Arg Lys Tyr Asn Ser Ala Pro Phe
85 90 95
Thr Phe Gly Gln Gly Thr Arg Val Glu Ile Lys
100 105
<210> 150
<211> 107
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VL (VK) S309-v12 mAb SARS-CoV-2
<400> 150
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Thr Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Asn Asn Tyr
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Lys Leu Leu Ile
35 40 45
Tyr Gly Ala Ser Thr Leu Gln Ser Gly Val Pro Ser Arg Phe Arg Gly
50 55 60
Ser Gly Ser Gly Thr Gly Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Val Ala Ser Tyr Tyr Cys Arg Lys Tyr Asn Ser Ala Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Arg Val Glu Ile Lys
100 105
<210> 151
<211> 9
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRL3 S309-v9 mAb SARS-CoV-2
<400> 151
Arg Lys Tyr Asn Ser Ala Pro Gly Thr
1 5
<210> 152
<211> 9
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRL3 S309-v10 mAb SARS-CoV-2
<400> 152
Arg Lys Tyr Asn Ser Ala Pro Arg Thr
1 5
<210> 153
<211> 9
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRL3 S309-v11 mAb SARS-CoV-2
<400> 153
Arg Lys Tyr Asn Ser Ala Pro Phe Thr
1 5
<210> 154
<211> 9
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRL3 S309-v12 mAb SARS-CoV-2
<400> 154
Arg Lys Tyr Asn Ser Ala Pro Tyr Thr
1 5
<210> 155
<211> 126
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S310 mAb SARS-CoV-2
<400> 155
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Leu Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Asn Ser Tyr
20 25 30
Ser Phe Asn Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Leu
35 40 45
Gly Gly Ile Ile Pro Val Leu Gly Thr Ser Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Ala Val Thr Ala Asp Glu Phe Thr Thr Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Arg Thr Tyr Asp Ser Ser Gly Tyr Arg Pro Tyr Tyr Tyr Gly
100 105 110
Leu Asp Val Trp Gly Gln Gly Thr Pro Val Thr Val Ser Ser
115 120 125
<210> 156
<211> 8
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH1 S310 mAb SARS-CoV-2
<400> 156
Gly Gly Thr Phe Asn Ser Tyr Ser
1 5
<210> 157
<211> 8
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH2 S310 mAb SARS-CoV-2
<400> 157
Ile Ile Pro Val Leu Gly Thr Ser
1 5
<210> 158
<211> 19
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH3 S310 mAb SARS-CoV-2
<400> 158
Ala Thr Arg Thr Tyr Asp Ser Ser Gly Tyr Arg Pro Tyr Tyr Tyr Gly
1 5 10 15
Leu Asp Val
<210> 159
<211> 110
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VL (VK) S310 mAb SARS-CoV-2
<400> 159
Gln Ser Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Ile Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Ser Tyr
20 25 30
Asn Leu Val Ser Trp Tyr Gln Gln Arg Pro Gly Lys Ala Pro Glu Leu
35 40 45
Met Ile Tyr Glu Val Thr Lys Arg Pro Ser Gly Leu Ser Asn Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Cys Ser Tyr Ala Gly Ser
85 90 95
Asp Thr Val Ile Phe Gly Gly Gly Thr Lys Val Thr Val Leu
100 105 110
<210> 160
<211> 9
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRL1 S310 mAb SARS-CoV-2
<400> 160
Ser Ser Asp Val Gly Ser Tyr Asn Leu
1 5
<210> 161
<211> 3
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRL2 S310 mAb SARS-CoV-2
<400> 161
Glu Val Thr
1
<210> 162
<211> 10
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRL3 S310 mAb SARS-CoV-2
<400> 162
Cys Ser Tyr Ala Gly Ser Asp Thr Val Ile
1 5 10
<210> 163
<211> 29902
<212> ДНК
<213> бетакоронавирус SARS коронавирус 2
<400> 163
attaaaggtt tataccttcc caggtaacaa accaaccaac tttcgatctc ttgtagatct 60
gttctctaaa cgaactttaa aatctgtgtg gctgtcactc ggctgcatgc ttagtgcact 120
cacgcagtat aattaataac taattactgt cgttgacagg acacgagtaa ctcgtctatc 180
ttctgcaggc tgcttacggt ttcgtccgtg ttgcagccga tcatcagcac atctaggttt 240
cgtccgggtg tgaccgaaag gtaagatgga gagccttgtc cctggtttca acgagaaaac 300
acacgtccaa ctcagtttgc ctgttttaca ggttcgcgac gtgctcgtac gtggctttgg 360
agactccgtg gaggaggtct tatcagaggc cgtcaacatc ttaaagatgg cacttgtggc 420
ttagtagaag ttgaaaaagg cgttttgcct caacttgaac agccctatgt gttcatcaaa 480
cgttcggatg ctcgaactgc acctcatggt catgttatgg ttgagctggt agcagaactc 540
gaaggcattc agtacggtcg tagtggtgag acacttggtg tccttgtccc tcatgtgggc 600
gaaataccag tggcttaccg caaggttctt cttcgtaaga acggtaataa aggagctggt 660
ggccatagtt acggcgccga tctaaagtca tttgacttag gcgacgagct tggcactgat 720
ccttatgaag attttcaaga aaactggaac actaaacata gcagtggtgt tacccgtgaa 780
ctcatgcgtg agcttaacgg aggggcatac actcgctatg tcgataacaa cttctgtggc 840
cctgatggct accctcttga gtgcattaaa gaccttctag cacgtgctgg taaagcttca 900
tgcactttgt ccgaacaact ggactttatt gacactaaga ggggtgtata ctgctgccgt 960
gaacatgagc atgaaattgc ttggtacacg gaacgttctg aaaagagcta tgaattgcag 1020
acaccttttg aaattaaatt ggcaaagaaa tttgacacct tcaatgggga atgtccaaat 1080
tttgtatttc ccttaaattc cataatcaag actattcaac caagggttga aaagaaaaag 1140
cttgatggct ttatgggtag aattcgatct gtctatccag ttgcgtcacc aaatgaatgc 1200
aaccaaatgt gcctttcaac tctcatgaag tgtgatcatt gtggtgaaac ttcatggcag 1260
acgggcgatt ttgttaaagc cacttgcgaa ttttgtggca ctgagaattt gactaaagaa 1320
ggtgccacta cttgtggtta cttaccccaa aatgctgttg ttaaaattta ttgtccagca 1380
tgtcacaatt cagaagtagg acctgagcat agtcttgccg aataccataa tgaatctggc 1440
ttgaaaacca ttcttcgtaa gggtggtcgc actattgcct ttggaggctg tgtgttctct 1500
tatgttggtt gccataacaa gtgtgcctat tgggttccac gtgctagcgc taacataggt 1560
tgtaaccata caggtgttgt tggagaaggt tccgaaggtc ttaatgacaa ccttcttgaa 1620
atactccaaa aagagaaagt caacatcaat attgttggtg actttaaact taatgaagag 1680
atcgccatta ttttggcatc tttttctgct tccacaagtg cttttgtgga aactgtgaaa 1740
ggtttggatt ataaagcatt caaacaaatt gttgaatcct gtggtaattt taaagttaca 1800
aaaggaaaag ctaaaaaagg tgcctggaat attggtgaac agaaatcaat actgagtcct 1860
ctttatgcat ttgcatcaga ggctgctcgt gttgtacgat caattttctc ccgcactctt 1920
gaaactgctc aaaattctgt gcgtgtttta cagaaggccg ctataacaat actagatgga 1980
atttcacagt attcactgag actcattgat gctatgatgt tcacatctga tttggctact 2040
aacaatctag ttgtaatggc ctacattaca ggtggtgttg ttcagttgac ttcgcagtgg 2100
ctaactaaca tctttggcac tgtttatgaa aaactcaaac ccgtccttga ttggcttgaa 2160
gagaagttta aggaaggtgt agagtttctt agagacggtt gggaaattgt taaatttatc 2220
tcaacctgtg cttgtgaaat tgtcggtgga caaattgtca cctgtgcaaa ggaaattaag 2280
gagagtgttc agacattctt taagcttgta aataaatttt tggctttgtg tgctgactct 2340
atcattattg gtggagctaa acttaaagcc ttgaatttag gtgaaacatt tgtcacgcac 2400
tcaaagggat tgtacagaaa gtgtgttaaa tccagagaag aaactggcct actcatgcct 2460
ctaaaagccc caaaagaaat tatcttctta gagggagaaa cacttcccac agaagtgtta 2520
acagaggaag ttgtcttgaa aactggtgat ttacaaccat tagaacaacc tactagtgaa 2580
gctgttgaag ctccattggt tggtacacca gtttgtatta acgggcttat gttgctcgaa 2640
atcaaagaca cagaaaagta ctgtgccctt gcacctaata tgatggtaac aaacaatacc 2700
ttcacactca aaggcggtgc accaacaaag gttacttttg gtgatgacac tgtgatagaa 2760
gtgcaaggtt acaagagtgt gaatatcact tttgaacttg atgaaaggat tgataaagta 2820
cttaatgaga agtgctctgc ctatacagtt gaactcggta cagaagtaaa tgagttcgcc 2880
tgtgttgtgg cagatgctgt cataaaaact ttgcaaccag tatctgaatt acttacacca 2940
ctgggcattg atttagatga gtggagtatg gctacatact acttatttga tgagtctggt 3000
gagtttaaat tggcttcaca tatgtattgt tctttctacc ctccagatga ggatgaagaa 3060
gaaggtgatt gtgaagaaga agagtttgag ccatcaactc aatatgagta tggtactgaa 3120
gatgattacc aaggtaaacc tttggaattt ggtgccactt ctgctgctct tcaacctgaa 3180
gaagagcaag aagaagattg gttagatgat gatagtcaac aaactgttgg tcaacaagac 3240
ggcagtgagg acaatcagac aactactatt caaacaattg ttgaggttca acctcaatta 3300
gagatggaac ttacaccagt tgttcagact attgaagtga atagttttag tggttattta 3360
aaacttactg acaatgtata cattaaaaat gcagacattg tggaagaagc taaaaaggta 3420
aaaccaacag tggttgttaa tgcagccaat gtttacctta aacatggagg aggtgttgca 3480
ggagccttaa ataaggctac taacaatgcc atgcaagttg aatctgatga ttacatagct 3540
actaatggac cacttaaagt gggtggtagt tgtgttttaa gcggacacaa tcttgctaaa 3600
cactgtcttc atgttgtcgg cccaaatgtt aacaaaggtg aagacattca acttcttaag 3660
agtgcttatg aaaattttaa tcagcacgaa gttctacttg caccattatt atcagctggt 3720
atttttggtg ctgaccctat acattcttta agagtttgtg tagatactgt tcgcacaaat 3780
gtctacttag ctgtctttga taaaaatctc tatgacaaac ttgtttcaag ctttttggaa 3840
atgaagagtg aaaagcaagt tgaacaaaag atcgctgaga ttcctaaaga ggaagttaag 3900
ccatttataa ctgaaagtaa accttcagtt gaacagagaa aacaagatga taagaaaatc 3960
aaagcttgtg ttgaagaagt tacaacaact ctggaagaaa ctaagttcct cacagaaaac 4020
ttgttacttt atattgacat taatggcaat cttcatccag attctgccac tcttgttagt 4080
gacattgaca tcactttctt aaagaaagat gctccatata tagtgggtga tgttgttcaa 4140
gagggtgttt taactgctgt ggttatacct actaaaaagg ctggtggcac tactgaaatg 4200
ctagcgaaag ctttgagaaa agtgccaaca gacaattata taaccactta cccgggtcag 4260
ggtttaaatg gttacactgt agaggaggca aagacagtgc ttaaaaagtg taaaagtgcc 4320
ttttacattc taccatctat tatctctaat gagaagcaag aaattcttgg aactgtttct 4380
tggaatttgc gagaaatgct tgcacatgca gaagaaacac gcaaattaat gcctgtctgt 4440
gtggaaacta aagccatagt ttcaactata cagcgtaaat ataagggtat taaaatacaa 4500
gagggtgtgg ttgattatgg tgctagattt tacttttaca ccagtaaaac aactgtagcg 4560
tcacttatca acacacttaa cgatctaaat gaaactcttg ttacaatgcc acttggctat 4620
gtaacacatg gcttaaattt ggaagaagct gctcggtata tgagatctct caaagtgcca 4680
gctacagttt ctgtttcttc acctgatgct gttacagcgt ataatggtta tcttacttct 4740
tcttctaaaa cacctgaaga acattttatt gaaaccatct cacttgctgg ttcctataaa 4800
gattggtcct attctggaca atctacacaa ctaggtatag aatttcttaa gagaggtgat 4860
aaaagtgtat attacactag taatcctacc acattccacc tagatggtga agttatcacc 4920
tttgacaatc ttaagacact tctttctttg agagaagtga ggactattaa ggtgtttaca 4980
acagtagaca acattaacct ccacacgcaa gttgtggaca tgtcaatgac atatggacaa 5040
cagtttggtc caacttattt ggatggagct gatgttacta aaataaaacc tcataattca 5100
catgaaggta aaacatttta tgttttacct aatgatgaca ctctacgtgt tgaggctttt 5160
gagtactacc acacaactga tcctagtttt ctgggtaggt acatgtcagc attaaatcac 5220
actaaaaagt ggaaataccc acaagttaat ggtttaactt ctattaaatg ggcagataac 5280
aactgttatc ttgccactgc attgttaaca ctccaacaaa tagagttgaa gtttaatcca 5340
cctgctctac aagatgctta ttacagagca agggctggtg aagctgctaa cttttgtgca 5400
cttatcttag cctactgtaa taagacagta ggtgagttag gtgatgttag agaaacaatg 5460
agttacttgt ttcaacatgc caatttagat tcttgcaaaa gagtcttgaa cgtggtgtgt 5520
aaaacttgtg gacaacagca gacaaccctt aagggtgtag aagctgttat gtacatgggc 5580
acactttctt atgaacaatt taagaaaggt gttcagatac cttgtacgtg tggtaaacaa 5640
gctacaaaat atctagtaca acaggagtca ccttttgtta tgatgtcagc accacctgct 5700
cagtatgaac ttaagcatgg tacatttact tgtgctagtg agtacactgg taattaccag 5760
tgtggtcact ataaacatat aacttctaaa gaaactttgt attgcataga cggtgcttta 5820
cttacaaagt cctcagaata caaaggtcct attacggatg ttttctacaa agaaaacagt 5880
tacacaacaa ccataaaacc agttacttat aaattggatg gtgttgtttg tacagaaatt 5940
gaccctaagt tggacaatta ttataagaaa gacaattctt atttcacaga gcaaccaatt 6000
gatcttgtac caaaccaacc atatccaaac gcaagcttcg ataattttaa gtttgtatgt 6060
gataatatca aatttgctga tgatttaaac cagttaactg gttataagaa acctgcttca 6120
agagagctta aagttacatt tttccctgac ttaaatggtg atgtggtggc tattgattat 6180
aaacactaca caccctcttt taagaaagga gctaaattgt tacataaacc tattgtttgg 6240
catgttaaca atgcaactaa taaagccacg tataaaccaa atacctggtg tatacgttgt 6300
ctttggagca caaaaccagt tgaaacatca aattcgtttg atgtactgaa gtcagaggac 6360
gcgcagggaa tggataatct tgcctgcgaa gatctaaaac cagtctctga agaagtagtg 6420
gaaaatccta ccatacagaa agacgttctt gagtgtaatg tgaaaactac cgaagttgta 6480
ggagacatta tacttaaacc agcaaataat agtttaaaaa ttacagaaga ggttggccac 6540
acagatctaa tggctgctta tgtagacaat tctagtctta ctattaagaa acctaatgaa 6600
ttatctagag tattaggttt gaaaaccctt gctactcatg gtttagctgc tgttaatagt 6660
gtcccttggg atactatagc taattatgct aagccttttc ttaacaaagt tgttagtaca 6720
actactaaca tagttacacg gtgtttaaac cgtgtttgta ctaattatat gccttatttc 6780
tttactttat tgctacaatt gtgtactttt actagaagta caaattctag aattaaagca 6840
tctatgccga ctactatagc aaagaatact gttaagagtg tcggtaaatt ttgtctagag 6900
gcttcattta attatttgaa gtcacctaat ttttctaaac tgataaatat tataatttgg 6960
tttttactat taagtgtttg cctaggttct ttaatctact caaccgctgc tttaggtgtt 7020
ttaatgtcta atttaggcat gccttcttac tgtactggtt acagagaagg ctatttgaac 7080
tctactaatg tcactattgc aacctactgt actggttcta taccttgtag tgtttgtctt 7140
agtggtttag attctttaga cacctatcct tctttagaaa ctatacaaat taccatttca 7200
tcttttaaat gggatttaac tgcttttggc ttagttgcag agtggttttt ggcatatatt 7260
cttttcacta ggtttttcta tgtacttgga ttggctgcaa tcatgcaatt gtttttcagc 7320
tattttgcag tacattttat tagtaattct tggcttatgt ggttaataat taatcttgta 7380
caaatggccc cgatttcagc tatggttaga atgtacatct tctttgcatc attttattat 7440
gtatggaaaa gttatgtgca tgttgtagac ggttgtaatt catcaacttg tatgatgtgt 7500
tacaaacgta atagagcaac aagagtcgaa tgtacaacta ttgttaatgg tgttagaagg 7560
tccttttatg tctatgctaa tggaggtaaa ggcttttgca aactacacaa ttggaattgt 7620
gttaattgtg atacattctg tgctggtagt acatttatta gtgatgaagt tgcgagagac 7680
ttgtcactac agtttaaaag accaataaat cctactgacc agtcttctta catcgttgat 7740
agtgttacag tgaagaatgg ttccatccat ctttactttg ataaagctgg tcaaaagact 7800
tatgaaagac attctctctc tcattttgtt aacttagaca acctgagagc taataacact 7860
aaaggttcat tgcctattaa tgttatagtt tttgatggta aatcaaaatg tgaagaatca 7920
tctgcaaaat cagcgtctgt ttactacagt cagcttatgt gtcaacctat actgttacta 7980
gatcaggcat tagtgtctga tgttggtgat agtgcggaag ttgcagttaa aatgtttgat 8040
gcttacgtta atacgttttc atcaactttt aacgtaccaa tggaaaaact caaaacacta 8100
gttgcaactg cagaagctga acttgcaaag aatgtgtcct tagacaatgt cttatctact 8160
tttatttcag cagctcggca agggtttgtt gattcagatg tagaaactaa agatgttgtt 8220
gaatgtctta aattgtcaca tcaatctgac atagaagtta ctggcgatag ttgtaataac 8280
tatatgctca cctataacaa agttgaaaac atgacacccc gtgaccttgg tgcttgtatt 8340
gactgtagtg cgcgtcatat taatgcgcag gtagcaaaaa gtcacaacat tgctttgata 8400
tggaacgtta aagatttcat gtcattgtct gaacaactac gaaaacaaat acgtagtgct 8460
gctaaaaaga ataacttacc ttttaagttg acatgtgcaa ctactagaca agttgttaat 8520
gttgtaacaa caaagatagc acttaagggt ggtaaaattg ttaataattg gttgaagcag 8580
ttaattaaag ttacacttgt gttccttttt gttgctgcta ttttctattt aataacacct 8640
gttcatgtca tgtctaaaca tactgacttt tcaagtgaaa tcataggata caaggctatt 8700
gatggtggtg tcactcgtga catagcatct acagatactt gttttgctaa caaacatgct 8760
gattttgaca catggtttag ccagcgtggt ggtagttata ctaatgacaa agcttgccca 8820
ttgattgctg cagtcataac aagagaagtg ggttttgtcg tgcctggttt gcctggcacg 8880
atattacgca caactaatgg tgactttttg catttcttac ctagagtttt tagtgcagtt 8940
ggtaacatct gttacacacc atcaaaactt atagagtaca ctgactttgc aacatcagct 9000
tgtgttttgg ctgctgaatg tacaattttt aaagatgctt ctggtaagcc agtaccatat 9060
tgttatgata ccaatgtact agaaggttct gttgcttatg aaagtttacg ccctgacaca 9120
cgttatgtgc tcatggatgg ctctattatt caatttccta acacctacct tgaaggttct 9180
gttagagtgg taacaacttt tgattctgag tactgtaggc acggcacttg tgaaagatca 9240
gaagctggtg tttgtgtatc tactagtggt agatgggtac ttaacaatga ttattacaga 9300
tctttaccag gagttttctg tggtgtagat gctgtaaatt tacttactaa tatgtttaca 9360
ccactaattc aacctattgg tgctttggac atatcagcat ctatagtagc tggtggtatt 9420
gtagctatcg tagtaacatg ccttgcctac tattttatga ggtttagaag agcttttggt 9480
gaatacagtc atgtagttgc ctttaatact ttactattcc ttatgtcatt cactgtactc 9540
tgtttaacac cagtttactc attcttacct ggtgtttatt ctgttattta cttgtacttg 9600
acattttatc ttactaatga tgtttctttt ttagcacata ttcagtggat ggttatgttc 9660
acacctttag tacctttctg gataacaatt gcttatatca tttgtatttc cacaaagcat 9720
ttctattggt tctttagtaa ttacctaaag agacgtgtag tctttaatgg tgtttccttt 9780
agtacttttg aagaagctgc gctgtgcacc tttttgttaa ataaagaaat gtatctaaag 9840
ttgcgtagtg atgtgctatt acctcttacg caatataata gatacttagc tctttataat 9900
aagtacaagt attttagtgg agcaatggat acaactagct acagagaagc tgcttgttgt 9960
catctcgcaa aggctctcaa tgacttcagt aactcaggtt ctgatgttct ttaccaacca 10020
ccacaaacct ctatcacctc agctgttttg cagagtggtt ttagaaaaat ggcattccca 10080
tctggtaaag ttgagggttg tatggtacaa gtaacttgtg gtacaactac acttaacggt 10140
ctttggcttg atgacgtagt ttactgtcca agacatgtga tctgcacctc tgaagacatg 10200
cttaacccta attatgaaga tttactcatt cgtaagtcta atcataattt cttggtacag 10260
gctggtaatg ttcaactcag ggttattgga cattctatgc aaaattgtgt acttaagctt 10320
aaggttgata cagccaatcc taagacacct aagtataagt ttgttcgcat tcaaccagga 10380
cagacttttt cagtgttagc ttgttacaat ggttcaccat ctggtgttta ccaatgtgct 10440
atgaggccca atttcactat taagggttca ttccttaatg gttcatgtgg tagtgttggt 10500
tttaacatag attatgactg tgtctctttt tgttacatgc accatatgga attaccaact 10560
ggagttcatg ctggcacaga cttagaaggt aacttttatg gaccttttgt tgacaggcaa 10620
acagcacaag cagctggtac ggacacaact attacagtta atgttttagc ttggttgtac 10680
gctgctgtta taaatggaga caggtggttt ctcaatcgat ttaccacaac tcttaatgac 10740
tttaaccttg tggctatgaa gtacaattat gaacctctaa cacaagacca tgttgacata 10800
ctaggacctc tttctgctca aactggaatt gccgttttag atatgtgtgc ttcattaaaa 10860
gaattactgc aaaatggtat gaatggacgt accatattgg gtagtgcttt attagaagat 10920
gaatttacac cttttgatgt tgttagacaa tgctcaggtg ttactttcca aagtgcagtg 10980
aaaagaacaa tcaagggtac acaccactgg ttgttactca caattttgac ttcactttta 11040
gttttagtcc agagtactca atggtctttg ttcttttttt tgtatgaaaa tgccttttta 11100
ccttttgcta tgggtattat tgctatgtct gcttttgcaa tgatgtttgt caaacataag 11160
catgcatttc tctgtttgtt tttgttacct tctcttgcca ctgtagctta ttttaatatg 11220
gtctatatgc ctgctagttg ggtgatgcgt attatgacat ggttggatat ggttgatact 11280
agtttgtctg gttttaagct aaaagactgt gttatgtatg catcagctgt agtgttacta 11340
atccttatga cagcaagaac tgtgtatgat gatggtgcta ggagagtgtg gacacttatg 11400
aatgtcttga cactcgttta taaagtttat tatggtaatg ctttagatca agccatttcc 11460
atgtgggctc ttataatctc tgttacttct aactactcag gtgtagttac aactgtcatg 11520
tttttggcca gaggtattgt ttttatgtgt gttgagtatt gccctatttt cttcataact 11580
ggtaatacac ttcagtgtat aatgctagtt tattgtttct taggctattt ttgtacttgt 11640
tactttggcc tcttttgttt actcaaccgc tactttagac tgactcttgg tgtttatgat 11700
tacttagttt ctacacagga gtttagatat atgaattcac agggactact cccacccaag 11760
aatagcatag atgccttcaa actcaacatt aaattgttgg gtgttggtgg caaaccttgt 11820
atcaaagtag ccactgtaca gtctaaaatg tcagatgtaa agtgcacatc agtagtctta 11880
ctctcagttt tgcaacaact cagagtagaa tcatcatcta aattgtgggc tcaatgtgtc 11940
cagttacaca atgacattct cttagctaaa gatactactg aagcctttga aaaaatggtt 12000
tcactacttt ctgttttgct ttccatgcag ggtgctgtag acataaacaa gctttgtgaa 12060
gaaatgctgg acaacagggc aaccttacaa gctatagcct cagagtttag ttcccttcca 12120
tcatatgcag cttttgctac tgctcaagaa gcttatgagc aggctgttgc taatggtgat 12180
tctgaagttg ttcttaaaaa gttgaagaag tctttgaatg tggctaaatc tgaatttgac 12240
cgtgatgcag ccatgcaacg taagttggaa aagatggctg atcaagctat gacccaaatg 12300
tataaacagg ctagatctga ggacaagagg gcaaaagtta ctagtgctat gcagacaatg 12360
cttttcacta tgcttagaaa gttggataat gatgcactca acaacattat caacaatgca 12420
agagatggtt gtgttccctt gaacataata cctcttacaa cagcagccaa actaatggtt 12480
gtcataccag actataacac atataaaaat acgtgtgatg gtacaacatt tacttatgca 12540
tcagcattgt gggaaatcca acaggttgta gatgcagata gtaaaattgt tcaacttagt 12600
gaaattagta tggacaattc acctaattta gcatggcctc ttattgtaac agctttaagg 12660
gccaattctg ctgtcaaatt acagaataat gagcttagtc ctgttgcact acgacagatg 12720
tcttgtgctg ccggtactac acaaactgct tgcactgatg acaatgcgtt agcttactac 12780
aacacaacaa agggaggtag gtttgtactt gcactgttat ccgatttaca ggatttgaaa 12840
tgggctagat tccctaagag tgatggaact ggtactatct atacagaact ggaaccacct 12900
tgtaggtttg ttacagacac acctaaaggt cctaaagtga agtatttata ctttattaaa 12960
ggattaaaca acctaaatag aggtatggta cttggtagtt tagctgccac agtacgtcta 13020
caagctggta atgcaacaga agtgcctgcc aattcaactg tattatcttt ctgtgctttt 13080
gctgtagatg ctgctaaagc ttacaaagat tatctagcta gtgggggaca accaatcact 13140
aattgtgtta agatgttgtg tacacacact ggtactggtc aggcaataac agttacaccg 13200
gaagccaata tggatcaaga atcctttggt ggtgcatcgt gttgtctgta ctgccgttgc 13260
cacatagatc atccaaatcc taaaggattt tgtgacttaa aaggtaagta tgtacaaata 13320
cctacaactt gtgctaatga ccctgtgggt tttacactta aaaacacagt ctgtaccgtc 13380
tgcggtatgt ggaaaggtta tggctgtagt tgtgatcaac tccgcgaacc catgcttcag 13440
tcagctgatg cacaatcgtt tttaaacggg tttgcggtgt aagtgcagcc cgtcttacac 13500
cgtgcggcac aggcactagt actgatgtcg tatacagggc ttttgacatc tacaatgata 13560
aagtagctgg ttttgctaaa ttcctaaaaa ctaattgttg tcgcttccaa gaaaaggacg 13620
aagatgacaa tttaattgat tcttactttg tagttaagag acacactttc tctaactacc 13680
aacatgaaga aacaatttat aatttactta aggattgtcc agctgttgct aaacatgact 13740
tctttaagtt tagaatagac ggtgacatgg taccacatat atcacgtcaa cgtcttacta 13800
aatacacaat ggcagacctc gtctatgctt taaggcattt tgatgaaggt aattgtgaca 13860
cattaaaaga aatacttgtc acatacaatt gttgtgatga tgattatttc aataaaaagg 13920
actggtatga ttttgtagaa aacccagata tattacgcgt atacgccaac ttaggtgaac 13980
gtgtacgcca agctttgtta aaaacagtac aattctgtga tgccatgcga aatgctggta 14040
ttgttggtgt actgacatta gataatcaag atctcaatgg taactggtat gatttcggtg 14100
atttcataca aaccacgcca ggtagtggag ttcctgttgt agattcttat tattcattgt 14160
taatgcctat attaaccttg accagggctt taactgcaga gtcacatgtt gacactgact 14220
taacaaagcc ttacattaag tgggatttgt taaaatatga cttcacggaa gagaggttaa 14280
aactctttga ccgttatttt aaatattggg atcagacata ccacccaaat tgtgttaact 14340
gtttggatga cagatgcatt ctgcattgtg caaactttaa tgttttattc tctacagtgt 14400
tcccacctac aagttttgga ccactagtga gaaaaatatt tgttgatggt gttccatttg 14460
tagtttcaac tggataccac ttcagagagc taggtgttgt acataatcag gatgtaaact 14520
tacatagctc tagacttagt tttaaggaat tacttgtgta tgctgctgac cctgctatgc 14580
acgctgcttc tggtaatcta ttactagata aacgcactac gtgcttttca gtagctgcac 14640
ttactaacaa tgttgctttt caaactgtca aacccggtaa ttttaacaaa gacttctatg 14700
actttgctgt gtctaagggt ttctttaagg aaggaagttc tgttgaatta aaacacttct 14760
tctttgctca ggatggtaat gctgctatca gcgattatga ctactatcgt tataatctac 14820
caacaatgtg tgatatcaga caactactat ttgtagttga agttgttgat aagtactttg 14880
attgttacga tggtggctgt attaatgcta accaagtcat cgtcaacaac ctagacaaat 14940
cagctggttt tccatttaat aaatggggta aggctagact ttattatgat tcaatgagtt 15000
atgaggatca agatgcactt ttcgcatata caaaacgtaa tgtcatccct actataactc 15060
aaatgaatct taagtatgcc attagtgcaa agaatagagc tcgcaccgta gctggtgtct 15120
ctatctgtag tactatgacc aatagacagt ttcatcaaaa attattgaaa tcaatagccg 15180
ccactagagg agctactgta gtaattggaa caagcaaatt ctatggtggt tggcacaaca 15240
tgttaaaaac tgtttatagt gatgtagaaa accctcacct tatgggttgg gattatccta 15300
aatgtgatag agccatgcct aacatgctta gaattatggc ctcacttgtt cttgctcgca 15360
aacatacaac gtgttgtagc ttgtcacacc gtttctatag attagctaat gagtgtgctc 15420
aagtattgag tgaaatggtc atgtgtggcg gttcactata tgttaaacca ggtggaacct 15480
catcaggaga tgccacaact gcttatgcta atagtgtttt taacatttgt caagctgtca 15540
cggccaatgt taatgcactt ttatctactg atggtaacaa aattgccgat aagtatgtcc 15600
gcaatttaca acacagactt tatgagtgtc tctatagaaa tagagatgtt gacacagact 15660
ttgtgaatga gttttacgca tatttgcgta aacatttctc aatgatgata ctctctgacg 15720
atgctgttgt gtgtttcaat agcacttatg catctcaagg tctagtggct agcataaaga 15780
actttaagtc agttctttat tatcaaaaca atgtttttat gtctgaagca aaatgttgga 15840
ctgagactga ccttactaaa ggacctcatg aattttgctc tcaacataca atgctagtta 15900
aacagggtga tgattatgtg taccttcctt acccagatcc atcaagaatc ctaggggccg 15960
gctgttttgt agatgatatc gtaaaaacag atggtacact tatgattgaa cggttcgtgt 16020
ctttagctat agatgcttac ccacttacta aacatcctaa tcaggagtat gctgatgtct 16080
ttcatttgta cttacaatac ataagaaagc tacatgatga gttaacagga cacatgttag 16140
acatgtattc tgttatgctt actaatgata acacttcaag gtattgggaa cctgagtttt 16200
atgaggctat gtacacaccg catacagtct tacaggctgt tggggcttgt gttctttgca 16260
attcacagac ttcattaaga tgtggtgctt gcatacgtag accattctta tgttgtaaat 16320
gctgttacga ccatgtcata tcaacatcac ataaattagt cttgtctgtt aatccgtatg 16380
tttgcaatgc tccaggttgt gatgtcacag atgtgactca actttactta ggaggtatga 16440
gctattattg taaatcacat aaaccaccca ttagttttcc attgtgtgct aatggacaag 16500
tttttggttt atataaaaat acatgtgttg gtagcgataa tgttactgac tttaatgcaa 16560
ttgcaacatg tgactggaca aatgctggtg attacatttt agctaacacc tgtactgaaa 16620
gactcaagct ttttgcagca gaaacgctca aagctactga ggagacattt aaactgtctt 16680
atggtattgc tactgtacgt gaagtgctgt ctgacagaga attacatctt tcatgggaag 16740
ttggtaaacc tagaccacca cttaaccgaa attatgtctt tactggttat cgtgtaacta 16800
aaaacagtaa agtacaaata ggagagtaca cctttgaaaa aggtgactat ggtgatgctg 16860
ttgtttaccg aggtacaaca acttacaaat taaatgttgg tgattatttt gtgctgacat 16920
cacatacagt aatgccatta agtgcaccta cactagtgcc acaagagcac tatgttagaa 16980
ttactggctt atacccaaca ctcaatatct cagatgagtt ttctagcaat gttgcaaatt 17040
atcaaaaggt tggtatgcaa aagtattcta cactccaggg accacctggt actggtaaga 17100
gtcattttgc tattggccta gctctctact acccttctgc tcgcatagtg tatacagctt 17160
gctctcatgc cgctgttgat gcactatgtg agaaggcatt aaaatatttg cctatagata 17220
aatgtagtag aattatacct gcacgtgctc gtgtagagtg ttttgataaa ttcaaagtga 17280
attcaacatt agaacagtat gtcttttgta ctgtaaatgc attgcctgag acgacagcag 17340
atatagttgt ctttgatgaa atttcaatgg ccacaaatta tgatttgagt gttgtcaatg 17400
ccagattacg tgctaagcac tatgtgtaca ttggcgaccc tgctcaatta cctgcaccac 17460
gcacattgct aactaagggc acactagaac cagaatattt caattcagtg tgtagactta 17520
tgaaaactat aggtccagac atgttcctcg gaacttgtcg gcgttgtcct gctgaaattg 17580
ttgacactgt gagtgctttg gtttatgata ataagcttaa agcacataaa gacaaatcag 17640
ctcaatgctt taaaatgttt tataagggtg ttatcacgca tgatgtttca tctgcaatta 17700
acaggccaca aataggcgtg gtaagagaat tccttacacg taaccctgct tggagaaaag 17760
ctgtctttat ttcaccttat aattcacaga atgctgtagc ctcaaagatt ttgggactac 17820
caactcaaac tgttgattca tcacagggct cagaatatga ctatgtcata ttcactcaaa 17880
ccactgaaac agctcactct tgtaatgtaa acagatttaa tgttgctatt accagagcaa 17940
aagtaggcat actttgcata atgtctgata gagaccttta tgacaagttg caatttacaa 18000
gtcttgaaat tccacgtagg aatgtggcaa ctttacaagc tgaaaatgta acaggactct 18060
ttaaagattg tagtaaggta atcactgggt tacatcctac acaggcacct acacacctca 18120
gtgttgacac taaattcaaa actgaaggtt tatgtgttga catacctggc atacctaagg 18180
acatgaccta tagaagactc atctctatga tgggttttaa aatgaattat caagttaatg 18240
gttaccctaa catgtttatc acccgcgaag aagctataag acatgtacgt gcatggattg 18300
gcttcgatgt cgaggggtgt catgctacta gagaagctgt tggtaccaat ttacctttac 18360
agctaggttt ttctacaggt gttaacctag ttgctgtacc tacaggttat gttgatacac 18420
ctaataatac agatttttcc agagttagtg ctaaaccacc gcctggagat caatttaaac 18480
acctcatacc acttatgtac aaaggacttc cttggaatgt agtgcgtata aagattgtac 18540
aaatgttaag tgacacactt aaaaatctct ctgacagagt cgtatttgtc ttatgggcac 18600
atggctttga gttgacatct atgaagtatt ttgtgaaaat aggacctgag cgcacctgtt 18660
gtctatgtga tagacgtgcc acatgctttt ccactgcttc agacacttat gcctgttggc 18720
atcattctat tggatttgat tacgtctata atccgtttat gattgatgtt caacaatggg 18780
gttttacagg taacctacaa agcaaccatg atctgtattg tcaagtccat ggtaatgcac 18840
atgtagctag ttgtgatgca atcatgacta ggtgtctagc tgtccacgag tgctttgtta 18900
agcgtgttga ctggactatt gaatatccta taattggtga tgaactgaag attaatgcgg 18960
cttgtagaaa ggttcaacac atggttgtta aagctgcatt attagcagac aaattcccag 19020
ttcttcacga cattggtaac cctaaagcta ttaagtgtgt acctcaagct gatgtagaat 19080
ggaagttcta tgatgcacag ccttgtagtg acaaagctta taaaatagaa gaattattct 19140
attcttatgc cacacattct gacaaattca cagatggtgt atgcctattt tggaattgca 19200
atgtcgatag atatcctgct aattccattg tttgtagatt tgacactaga gtgctatcta 19260
accttaactt gcctggttgt gatggtggca gtttgtatgt aaataaacat gcattccaca 19320
caccagcttt tgataaaagt gcttttgtta atttaaaaca attaccattt ttctattact 19380
ctgacagtcc atgtgagtct catggaaaac aagtagtgtc agatatagat tatgtaccac 19440
taaagtctgc tacgtgtata acacgttgca atttaggtgg tgctgtctgt agacatcatg 19500
ctaatgagta cagattgtat ctcgatgctt ataacatgat gatctcagct ggctttagct 19560
tgtgggttta caaacaattt gatacttata acctctggaa cacttttaca agacttcaga 19620
gtttagaaaa tgtggctttt aatgttgtaa ataagggaca ctttgatgga caacagggtg 19680
aagtaccagt ttctatcatt aataacactg tttacacaaa agttgatggt gttgatgtag 19740
aattgtttga aaataaaaca acattacctg ttaatgtagc atttgagctt tgggctaagc 19800
gcaacattaa accagtacca gaggtgaaaa tactcaataa tttgggtgtg gacattgctg 19860
ctaatactgt gatctgggac tacaaaagag atgctccagc acatatatct actattggtg 19920
tttgttctat gactgacata gccaagaaac caactgaaac gatttgtgca ccactcactg 19980
tcttttttga tggtagagtt gatggtcaag tagacttatt tagaaatgcc cgtaatggtg 20040
ttcttattac agaaggtagt gttaaaggtt tacaaccatc tgtaggtccc aaacaagcta 20100
gtcttaatgg agtcacatta attggagaag ccgtaaaaac acagttcaat tattataaga 20160
aagttgatgg tgttgtccaa caattacctg aaacttactt tactcagagt agaaatttac 20220
aagaatttaa acccaggagt caaatggaaa ttgatttctt agaattagct atggatgaat 20280
tcattgaacg gtataaatta gaaggctatg ccttcgaaca tatcgtttat ggagatttta 20340
gtcatagtca gttaggtggt ttacatctac tgattggact agctaaacgt tttaaggaat 20400
caccttttga attagaagat tttattccta tggacagtac agttaaaaac tatttcataa 20460
cagatgcgca aacaggttca tctaagtgtg tgtgttctgt tattgattta ttacttgatg 20520
attttgttga aataataaaa tcccaagatt tatctgtagt ttctaaggtt gtcaaagtga 20580
ctattgacta tacagaaatt tcatttatgc tttggtgtaa agatggccat gtagaaacat 20640
tttacccaaa attacaatct agtcaagcgt ggcaaccggg tgttgctatg cctaatcttt 20700
acaaaatgca aagaatgcta ttagaaaagt gtgaccttca aaattatggt gatagtgcaa 20760
cattacctaa aggcataatg atgaatgtcg caaaatatac tcaactgtgt caatatttaa 20820
acacattaac attagctgta ccctataata tgagagttat acattttggt gctggttctg 20880
ataaaggagt tgcaccaggt acagctgttt taagacagtg gttgcctacg ggtacgctgc 20940
ttgtcgattc agatcttaat gactttgtct ctgatgcaga ttcaactttg attggtgatt 21000
gtgcaactgt acatacagct aataaatggg atctcattat tagtgatatg tacgacccta 21060
agactaaaaa tgttacaaaa gaaaatgact ctaaagaggg ttttttcact tacatttgtg 21120
ggtttataca acaaaagcta gctcttggag gttccgtggc tataaagata acagaacatt 21180
cttggaatgc tgatctttat aagctcatgg gacacttcgc atggtggaca gcctttgtta 21240
ctaatgtgaa tgcgtcatca tctgaagcat ttttaattgg atgtaattat cttggcaaac 21300
cacgcgaaca aatagatggt tatgtcatgc atgcaaatta catattttgg aggaatacaa 21360
atccaattca gttgtcttcc tattctttat ttgacatgag taaatttccc cttaaattaa 21420
ggggtactgc tgttatgtct ttaaaagaag gtcaaatcaa tgatatgatt ttatctcttc 21480
ttagtaaagg tagacttata attagagaaa acaacagagt tgttatttct agtgatgttc 21540
ttgttaacaa ctaaacgaac aatgtttgtt tttcttgttt tattgccact agtctctagt 21600
cagtgtgtta atcttacaac cagaactcaa ttaccccctg catacactaa ttctttcaca 21660
cgtggtgttt attaccctga caaagttttc agatcctcag ttttacattc aactcaggac 21720
ttgttcttac ctttcttttc caatgttact tggttccatg ctatacatgt ctctgggacc 21780
aatggtacta agaggtttga taaccctgtc ctaccattta atgatggtgt ttattttgct 21840
tccactgaga agtctaacat aataagaggc tggatttttg gtactacttt agattcgaag 21900
acccagtccc tacttattgt taataacgct actaatgttg ttattaaagt ctgtgaattt 21960
caattttgta atgatccatt tttgggtgtt tattaccaca aaaacaacaa aagttggatg 22020
gaaagtgagt tcagagttta ttctagtgcg aataattgca cttttgaata tgtctctcag 22080
ccttttctta tggaccttga aggaaaacag ggtaatttca aaaatcttag ggaatttgtg 22140
tttaagaata ttgatggtta ttttaaaata tattctaagc acacgcctat taatttagtg 22200
cgtgatctcc ctcagggttt ttcggcttta gaaccattgg tagatttgcc aataggtatt 22260
aacatcacta ggtttcaaac tttacttgct ttacatagaa gttatttgac tcctggtgat 22320
tcttcttcag gttggacagc tggtgctgca gcttattatg tgggttatct tcaacctagg 22380
acttttctat taaaatataa tgaaaatgga accattacag atgctgtaga ctgtgcactt 22440
gaccctctct cagaaacaaa gtgtacgttg aaatccttca ctgtagaaaa aggaatctat 22500
caaacttcta actttagagt ccaaccaaca gaatctattg ttagatttcc taatattaca 22560
aacttgtgcc cttttggtga agtttttaac gccaccagat ttgcatctgt ttatgcttgg 22620
aacaggaaga gaatcagcaa ctgtgttgct gattattctg tcctatataa ttccgcatca 22680
ttttccactt ttaagtgtta tggagtgtct cctactaaat taaatgatct ctgctttact 22740
aatgtctatg cagattcatt tgtaattaga ggtgatgaag tcagacaaat cgctccaggg 22800
caaactggaa agattgctga ttataattat aaattaccag atgattttac aggctgcgtt 22860
atagcttgga attctaacaa tcttgattct aaggttggtg gtaattataa ttacctgtat 22920
agattgttta ggaagtctaa tctcaaacct tttgagagag atatttcaac tgaaatctat 22980
caggccggta gcacaccttg taatggtgtt gaaggtttta attgttactt tcctttacaa 23040
tcatatggtt tccaacccac taatggtgtt ggttaccaac catacagagt agtagtactt 23100
tcttttgaac ttctacatgc accagcaact gtttgtggac ctaaaaagtc tactaatttg 23160
gttaaaaaca aatgtgtcaa tttcaacttc aatggtttaa caggcacagg tgttcttact 23220
gagtctaaca aaaagtttct gcctttccaa caatttggca gagacattgc tgacactact 23280
gatgctgtcc gtgatccaca gacacttgag attcttgaca ttacaccatg ttcttttggt 23340
ggtgtcagtg ttataacacc aggaacaaat acttctaacc aggttgctgt tctttatcag 23400
gatgttaact gcacagaagt ccctgttgct attcatgcag atcaacttac tcctacttgg 23460
cgtgtttatt ctacaggttc taatgttttt caaacacgtg caggctgttt aataggggct 23520
gaacatgtca acaactcata tgagtgtgac atacccattg gtgcaggtat atgcgctagt 23580
tatcagactc agactaattc tcctcggcgg gcacgtagtg tagctagtca atccatcatt 23640
gcctacacta tgtcacttgg tgcagaaaat tcagttgctt actctaataa ctctattgcc 23700
atacccacaa attttactat tagtgttacc acagaaattc taccagtgtc tatgaccaag 23760
acatcagtag attgtacaat gtacatttgt ggtgattcaa ctgaatgcag caatcttttg 23820
ttgcaatatg gcagtttttg tacacaatta aaccgtgctt taactggaat agctgttgaa 23880
caagacaaaa acacccaaga agtttttgca caagtcaaac aaatttacaa aacaccacca 23940
attaaagatt ttggtggttt taatttttca caaatattac cagatccatc aaaaccaagc 24000
aagaggtcat ttattgaaga tctacttttc aacaaagtga cacttgcaga tgctggcttc 24060
atcaaacaat atggtgattg ccttggtgat attgctgcta gagacctcat ttgtgcacaa 24120
aagtttaacg gccttactgt tttgccacct ttgctcacag atgaaatgat tgctcaatac 24180
acttctgcac tgttagcggg tacaatcact tctggttgga cctttggtgc aggtgctgca 24240
ttacaaatac catttgctat gcaaatggct tataggttta atggtattgg agttacacag 24300
aatgttctct atgagaacca aaaattgatt gccaaccaat ttaatagtgc tattggcaaa 24360
attcaagact cactttcttc cacagcaagt gcacttggaa aacttcaaga tgtggtcaac 24420
caaaatgcac aagctttaaa cacgcttgtt aaacaactta gctccaattt tggtgcaatt 24480
tcaagtgttt taaatgatat cctttcacgt cttgacaaag ttgaggctga agtgcaaatt 24540
gataggttga tcacaggcag acttcaaagt ttgcagacat atgtgactca acaattaatt 24600
agagctgcag aaatcagagc ttctgctaat cttgctgcta ctaaaatgtc agagtgtgta 24660
cttggacaat caaaaagagt tgatttttgt ggaaagggct atcatcttat gtccttccct 24720
cagtcagcac ctcatggtgt agtcttcttg catgtgactt atgtccctgc acaagaaaag 24780
aacttcacaa ctgctcctgc catttgtcat gatggaaaag cacactttcc tcgtgaaggt 24840
gtctttgttt caaatggcac acactggttt gtaacacaaa ggaattttta tgaaccacaa 24900
atcattacta cagacaacac atttgtgtct ggtaactgtg atgttgtaat aggaattgtc 24960
aacaacacag tttatgatcc tttgcaacct gaattagact cattcaagga ggagttagat 25020
aaatatttta agaatcatac atcaccagat gttgatttag gtgacatctc tggcattaat 25080
gcttcagttg taaacattca aaaagaaatt gaccgcctca atgaggttgc caagaattta 25140
aatgaatctc tcatcgatct ccaagaactt ggaaagtatg agcagtatat aaaatggcca 25200
tggtacattt ggctaggttt tatagctggc ttgattgcca tagtaatggt gacaattatg 25260
ctttgctgta tgaccagttg ctgtagttgt ctcaagggct gttgttcttg tggatcctgc 25320
tgcaaatttg atgaagacga ctctgagcca gtgctcaaag gagtcaaatt acattacaca 25380
taaacgaact tatggatttg tttatgagaa tcttcacaat tggaactgta actttgaagc 25440
aaggtgaaat caaggatgct actccttcag attttgttcg cgctactgca acgataccga 25500
tacaagcctc actccctttc ggatggctta ttgttggcgt tgcacttctt gctgtttttc 25560
agagcgcttc caaaatcata accctcaaaa agagatggca actagcactc tccaagggtg 25620
ttcactttgt ttgcaacttg ctgttgttgt ttgtaacagt ttactcacac cttttgctcg 25680
ttgctgctgg ccttgaagcc ccttttctct atctttatgc tttagtctac ttcttgcaga 25740
gtataaactt tgtaagaata ataatgaggc tttggctttg ctggaaatgc cgttccaaaa 25800
acccattact ttatgatgcc aactattttc tttgctggca tactaattgt tacgactatt 25860
gtatacctta caatagtgta acttcttcaa ttgtcattac ttcaggtgat ggcacaacaa 25920
gtcctatttc tgaacatgac taccagattg gtggttatac tgaaaaatgg gaatctggag 25980
taaaagactg tgttgtatta cacagttact tcacttcaga ctattaccag ctgtactcaa 26040
ctcaattgag tacagacact ggtgttgaac atgttacctt cttcatctac aataaaattg 26100
ttgatgagcc tgaagaacat gtccaaattc acacaatcga cggttcatcc ggagttgtta 26160
atccagtaat ggaaccaatt tatgatgaac cgacgacgac tactagcgtg cctttgtaag 26220
cacaagctga tgagtacgaa cttatgtact cattcgtttc ggaagagaca ggtacgttaa 26280
tagttaatag cgtacttctt tttcttgctt tcgtggtatt cttgctagtt acactagcca 26340
tccttactgc gcttcgattg tgtgcgtact gctgcaatat tgttaacgtg agtcttgtaa 26400
aaccttcttt ttacgtttac tctcgtgtta aaaatctgaa ttcttctaga gttcctgatc 26460
ttctggtcta aacgaactaa atattatatt agtttttctg tttggaactt taattttagc 26520
catggcagat tccaacggta ctattaccgt tgaagagctt aaaaagctcc ttgaacaatg 26580
gaacctagta ataggtttcc tattccttac atggatttgt cttctacaat ttgcctatgc 26640
caacaggaat aggtttttgt atataattaa gttaattttc ctctggctgt tatggccagt 26700
aactttagct tgttttgtgc ttgctgctgt ttacagaata aattggatca ccggtggaat 26760
tgctatcgca atggcttgtc ttgtaggctt gatgtggctc agctacttca ttgcttcttt 26820
cagactgttt gcgcgtacgc gttccatgtg gtcattcaat ccagaaacta acattcttct 26880
caacgtgcca ctccatggca ctattctgac cagaccgctt ctagaaagtg aactcgtaat 26940
cggagctgtg atccttcgtg gacatcttcg tattgctgga caccatctag gacgctgtga 27000
catcaaggac ctgcctaaag aaatcactgt tgctacatca cgaacgcttt cttattacaa 27060
attgggagct tcgcagcgtg tagcaggtga ctcaggtttt gctgcataca gtcgctacag 27120
gattggcaac tataaattaa acacagacca ttccagtagc agtgacaata ttgctttgct 27180
tgtacagtaa gtgacaacag atgtttcatc tcgttgactt tcaggttact atagcagaga 27240
tattactaat tattatgagg acttttaaag tttccatttg gaatcttgat tacatcataa 27300
acctcataat taaaaattta tctaagtcac taactgagaa taaatattct caattagatg 27360
aagagcaacc aatggagatt gattaaacga acatgaaaat tattcttttc ttggcactga 27420
taacactcgc tacttgtgag ctttatcact accaagagtg tgttagaggt acaacagtac 27480
ttttaaaaga accttgctct tctggaacat acgagggcaa ttcaccattt catcctctag 27540
ctgataacaa atttgcactg acttgcttta gcactcaatt tgcttttgct tgtcctgacg 27600
gcgtaaaaca cgtctatcag ttacgtgcca gatcagtttc acctaaactg ttcatcagac 27660
aagaggaagt tcaagaactt tactctccaa tttttcttat tgttgcggca atagtgttta 27720
taacactttg cttcacactc aaaagaaaga cagaatgatt gaactttcat taattgactt 27780
ctatttgtgc tttttagcct ttctgctatt ccttgtttta attatgctta ttatcttttg 27840
gttctcactt gaactgcaag atcataatga aacttgtcac gcctaaacga acatgaaatt 27900
tcttgttttc ttaggaatca tcacaactgt agctgcattt caccaagaat gtagtttaca 27960
gtcatgtact caacatcaac catatgtagt tgatgacccg tgtcctattc acttctattc 28020
taaatggtat attagagtag gagctagaaa atcagcacct ttaattgaat tgtgcgtgga 28080
tgaggctggt tctaaatcac ccattcagta catcgatatc ggtaattata cagtttcctg 28140
tttacctttt acaattaatt gccaggaacc taaattgggt agtcttgtag tgcgttgttc 28200
gttctatgaa gactttttag agtatcatga cgttcgtgtt gttttagatt tcatctaaac 28260
gaacaaacta aaatgtctga taatggaccc caaaatcagc gaaatgcacc ccgcattacg 28320
tttggtggac cctcagattc aactggcagt aaccagaatg gagaacgcag tggggcgcga 28380
tcaaaacaac gtcggcccca aggtttaccc aataatactg cgtcttggtt caccgctctc 28440
actcaacatg gcaaggaaga ccttaaattc cctcgaggac aaggcgttcc aattaacacc 28500
aatagcagtc cagatgacca aattggctac taccgaagag ctaccagacg aattcgtggt 28560
ggtgacggta aaatgaaaga tctcagtcca agatggtatt tctactacct aggaactggg 28620
ccagaagctg gacttcccta tggtgctaac aaagacggca tcatatgggt tgcaactgag 28680
ggagccttga atacaccaaa agatcacatt ggcacccgca atcctgctaa caatgctgca 28740
atcgtgctac aacttcctca aggaacaaca ttgccaaaag gcttctacgc agaagggagc 28800
agaggcggca gtcaagcctc ttctcgttcc tcatcacgta gtcgcaacag ttcaagaaat 28860
tcaactccag gcagcagtag gggaacttct cctgctagaa tggctggcaa tggcggtgat 28920
gctgctcttg ctttgctgct gcttgacaga ttgaaccagc ttgagagcaa aatgtctggt 28980
aaaggccaac aacaacaagg ccaaactgtc actaagaaat ctgctgctga ggcttctaag 29040
aagcctcggc aaaaacgtac tgccactaaa gcatacaatg taacacaagc tttcggcaga 29100
cgtggtccag aacaaaccca aggaaatttt ggggaccagg aactaatcag acaaggaact 29160
gattacaaac attggccgca aattgcacaa tttgccccca gcgcttcagc gttcttcgga 29220
atgtcgcgca ttggcatgga agtcacacct tcgggaacgt ggttgaccta cacaggtgcc 29280
atcaaattgg atgacaaaga tccaaatttc aaagatcaag tcattttgct gaataagcat 29340
attgacgcat acaaaacatt cccaccaaca gagcctaaaa aggacaaaaa gaagaaggct 29400
gatgaaactc aagccttacc gcagagacag aagaaacagc aaactgtgac tcttcttcct 29460
gctgcagatt tggatgattt ctccaaacaa ttgcaacaat ccatgagcag tgctgactca 29520
actcaggcct aaactcatgc agaccacaca aggcagatgg gctatataaa cgttttcgct 29580
tttccgttta cgatatatag tctactcttg tgcagaatga attctcgtaa ctacatagca 29640
caagtagatg tagttaactt taatctcaca tagcaatctt taatcagtgt gtaacattag 29700
ggaggacttg aaagagccac cacattttca ccgaggccac gcggagtacg atcgagtgta 29760
cagtgaacaa tgctagggag agctgcctat atggaagagc cctaatgtgt aaaattaatt 29820
ttagtagtgc tatccccatg tgattttaat agcttcttag gagaatgaca aaaaaaaaaa 29880
aaaaaaaaaa aaaaaaaaaa aa 29902
<210> 164
<211> 7096
<212> ПРТ
<213> бетакоронавирус SARS коронавирус 2
<400> 164
Met Glu Ser Leu Val Pro Gly Phe Asn Glu Lys Thr His Val Gln Leu
1 5 10 15
Ser Leu Pro Val Leu Gln Val Arg Asp Val Leu Val Arg Gly Phe Gly
20 25 30
Asp Ser Val Glu Glu Val Leu Ser Glu Ala Arg Gln His Leu Lys Asp
35 40 45
Gly Thr Cys Gly Leu Val Glu Val Glu Lys Gly Val Leu Pro Gln Leu
50 55 60
Glu Gln Pro Tyr Val Phe Ile Lys Arg Ser Asp Ala Arg Thr Ala Pro
65 70 75 80
His Gly His Val Met Val Glu Leu Val Ala Glu Leu Glu Gly Ile Gln
85 90 95
Tyr Gly Arg Ser Gly Glu Thr Leu Gly Val Leu Val Pro His Val Gly
100 105 110
Glu Ile Pro Val Ala Tyr Arg Lys Val Leu Leu Arg Lys Asn Gly Asn
115 120 125
Lys Gly Ala Gly Gly His Ser Tyr Gly Ala Asp Leu Lys Ser Phe Asp
130 135 140
Leu Gly Asp Glu Leu Gly Thr Asp Pro Tyr Glu Asp Phe Gln Glu Asn
145 150 155 160
Trp Asn Thr Lys His Ser Ser Gly Val Thr Arg Glu Leu Met Arg Glu
165 170 175
Leu Asn Gly Gly Ala Tyr Thr Arg Tyr Val Asp Asn Asn Phe Cys Gly
180 185 190
Pro Asp Gly Tyr Pro Leu Glu Cys Ile Lys Asp Leu Leu Ala Arg Ala
195 200 205
Gly Lys Ala Ser Cys Thr Leu Ser Glu Gln Leu Asp Phe Ile Asp Thr
210 215 220
Lys Arg Gly Val Tyr Cys Cys Arg Glu His Glu His Glu Ile Ala Trp
225 230 235 240
Tyr Thr Glu Arg Ser Glu Lys Ser Tyr Glu Leu Gln Thr Pro Phe Glu
245 250 255
Ile Lys Leu Ala Lys Lys Phe Asp Thr Phe Asn Gly Glu Cys Pro Asn
260 265 270
Phe Val Phe Pro Leu Asn Ser Ile Ile Lys Thr Ile Gln Pro Arg Val
275 280 285
Glu Lys Lys Lys Leu Asp Gly Phe Met Gly Arg Ile Arg Ser Val Tyr
290 295 300
Pro Val Ala Ser Pro Asn Glu Cys Asn Gln Met Cys Leu Ser Thr Leu
305 310 315 320
Met Lys Cys Asp His Cys Gly Glu Thr Ser Trp Gln Thr Gly Asp Phe
325 330 335
Val Lys Ala Thr Cys Glu Phe Cys Gly Thr Glu Asn Leu Thr Lys Glu
340 345 350
Gly Ala Thr Thr Cys Gly Tyr Leu Pro Gln Asn Ala Val Val Lys Ile
355 360 365
Tyr Cys Pro Ala Cys His Asn Ser Glu Val Gly Pro Glu His Ser Leu
370 375 380
Ala Glu Tyr His Asn Glu Ser Gly Leu Lys Thr Ile Leu Arg Lys Gly
385 390 395 400
Gly Arg Thr Ile Ala Phe Gly Gly Cys Val Phe Ser Tyr Val Gly Cys
405 410 415
His Asn Lys Cys Ala Tyr Trp Val Pro Arg Ala Ser Ala Asn Ile Gly
420 425 430
Cys Asn His Thr Gly Val Val Gly Glu Gly Ser Glu Gly Leu Asn Asp
435 440 445
Asn Leu Leu Glu Ile Leu Gln Lys Glu Lys Val Asn Ile Asn Ile Val
450 455 460
Gly Asp Phe Lys Leu Asn Glu Glu Ile Ala Ile Ile Leu Ala Ser Phe
465 470 475 480
Ser Ala Ser Thr Ser Ala Phe Val Glu Thr Val Lys Gly Leu Asp Tyr
485 490 495
Lys Ala Phe Lys Gln Ile Val Glu Ser Cys Gly Asn Phe Lys Val Thr
500 505 510
Lys Gly Lys Ala Lys Lys Gly Ala Trp Asn Ile Gly Glu Gln Lys Ser
515 520 525
Ile Leu Ser Pro Leu Tyr Ala Phe Ala Ser Glu Ala Ala Arg Val Val
530 535 540
Arg Ser Ile Phe Ser Arg Thr Leu Glu Thr Ala Gln Asn Ser Val Arg
545 550 555 560
Val Leu Gln Lys Ala Ala Ile Thr Ile Leu Asp Gly Ile Ser Gln Tyr
565 570 575
Ser Leu Arg Leu Ile Asp Ala Met Met Phe Thr Ser Asp Leu Ala Thr
580 585 590
Asn Asn Leu Val Val Met Ala Tyr Ile Thr Gly Gly Val Val Gln Leu
595 600 605
Thr Ser Gln Trp Leu Thr Asn Ile Phe Gly Thr Val Tyr Glu Lys Leu
610 615 620
Lys Pro Val Leu Asp Trp Leu Glu Glu Lys Phe Lys Glu Gly Val Glu
625 630 635 640
Phe Leu Arg Asp Gly Trp Glu Ile Val Lys Phe Ile Ser Thr Cys Ala
645 650 655
Cys Glu Ile Val Gly Gly Gln Ile Val Thr Cys Ala Lys Glu Ile Lys
660 665 670
Glu Ser Val Gln Thr Phe Phe Lys Leu Val Asn Lys Phe Leu Ala Leu
675 680 685
Cys Ala Asp Ser Ile Ile Ile Gly Gly Ala Lys Leu Lys Ala Leu Asn
690 695 700
Leu Gly Glu Thr Phe Val Thr His Ser Lys Gly Leu Tyr Arg Lys Cys
705 710 715 720
Val Lys Ser Arg Glu Glu Thr Gly Leu Leu Met Pro Leu Lys Ala Pro
725 730 735
Lys Glu Ile Ile Phe Leu Glu Gly Glu Thr Leu Pro Thr Glu Val Leu
740 745 750
Thr Glu Glu Val Val Leu Lys Thr Gly Asp Leu Gln Pro Leu Glu Gln
755 760 765
Pro Thr Ser Glu Ala Val Glu Ala Pro Leu Val Gly Thr Pro Val Cys
770 775 780
Ile Asn Gly Leu Met Leu Leu Glu Ile Lys Asp Thr Glu Lys Tyr Cys
785 790 795 800
Ala Leu Ala Pro Asn Met Met Val Thr Asn Asn Thr Phe Thr Leu Lys
805 810 815
Gly Gly Ala Pro Thr Lys Val Thr Phe Gly Asp Asp Thr Val Ile Glu
820 825 830
Val Gln Gly Tyr Lys Ser Val Asn Ile Thr Phe Glu Leu Asp Glu Arg
835 840 845
Ile Asp Lys Val Leu Asn Glu Lys Cys Ser Ala Tyr Thr Val Glu Leu
850 855 860
Gly Thr Glu Val Asn Glu Phe Ala Cys Val Val Ala Asp Ala Val Ile
865 870 875 880
Lys Thr Leu Gln Pro Val Ser Glu Leu Leu Thr Pro Leu Gly Ile Asp
885 890 895
Leu Asp Glu Trp Ser Met Ala Thr Tyr Tyr Leu Phe Asp Glu Ser Gly
900 905 910
Glu Phe Lys Leu Ala Ser His Met Tyr Cys Ser Phe Tyr Pro Pro Asp
915 920 925
Glu Asp Glu Glu Glu Gly Asp Cys Glu Glu Glu Glu Phe Glu Pro Ser
930 935 940
Thr Gln Tyr Glu Tyr Gly Thr Glu Asp Asp Tyr Gln Gly Lys Pro Leu
945 950 955 960
Glu Phe Gly Ala Thr Ser Ala Ala Leu Gln Pro Glu Glu Glu Gln Glu
965 970 975
Glu Asp Trp Leu Asp Asp Asp Ser Gln Gln Thr Val Gly Gln Gln Asp
980 985 990
Gly Ser Glu Asp Asn Gln Thr Thr Thr Ile Gln Thr Ile Val Glu Val
995 1000 1005
Gln Pro Gln Leu Glu Met Glu Leu Thr Pro Val Val Gln Thr Ile
1010 1015 1020
Glu Val Asn Ser Phe Ser Gly Tyr Leu Lys Leu Thr Asp Asn Val
1025 1030 1035
Tyr Ile Lys Asn Ala Asp Ile Val Glu Glu Ala Lys Lys Val Lys
1040 1045 1050
Pro Thr Val Val Val Asn Ala Ala Asn Val Tyr Leu Lys His Gly
1055 1060 1065
Gly Gly Val Ala Gly Ala Leu Asn Lys Ala Thr Asn Asn Ala Met
1070 1075 1080
Gln Val Glu Ser Asp Asp Tyr Ile Ala Thr Asn Gly Pro Leu Lys
1085 1090 1095
Val Gly Gly Ser Cys Val Leu Ser Gly His Asn Leu Ala Lys His
1100 1105 1110
Cys Leu His Val Val Gly Pro Asn Val Asn Lys Gly Glu Asp Ile
1115 1120 1125
Gln Leu Leu Lys Ser Ala Tyr Glu Asn Phe Asn Gln His Glu Val
1130 1135 1140
Leu Leu Ala Pro Leu Leu Ser Ala Gly Ile Phe Gly Ala Asp Pro
1145 1150 1155
Ile His Ser Leu Arg Val Cys Val Asp Thr Val Arg Thr Asn Val
1160 1165 1170
Tyr Leu Ala Val Phe Asp Lys Asn Leu Tyr Asp Lys Leu Val Ser
1175 1180 1185
Ser Phe Leu Glu Met Lys Ser Glu Lys Gln Val Glu Gln Lys Ile
1190 1195 1200
Ala Glu Ile Pro Lys Glu Glu Val Lys Pro Phe Ile Thr Glu Ser
1205 1210 1215
Lys Pro Ser Val Glu Gln Arg Lys Gln Asp Asp Lys Lys Ile Lys
1220 1225 1230
Ala Cys Val Glu Glu Val Thr Thr Thr Leu Glu Glu Thr Lys Phe
1235 1240 1245
Leu Thr Glu Asn Leu Leu Leu Tyr Ile Asp Ile Asn Gly Asn Leu
1250 1255 1260
His Pro Asp Ser Ala Thr Leu Val Ser Asp Ile Asp Ile Thr Phe
1265 1270 1275
Leu Lys Lys Asp Ala Pro Tyr Ile Val Gly Asp Val Val Gln Glu
1280 1285 1290
Gly Val Leu Thr Ala Val Val Ile Pro Thr Lys Lys Ala Gly Gly
1295 1300 1305
Thr Thr Glu Met Leu Ala Lys Ala Leu Arg Lys Val Pro Thr Asp
1310 1315 1320
Asn Tyr Ile Thr Thr Tyr Pro Gly Gln Gly Leu Asn Gly Tyr Thr
1325 1330 1335
Val Glu Glu Ala Lys Thr Val Leu Lys Lys Cys Lys Ser Ala Phe
1340 1345 1350
Tyr Ile Leu Pro Ser Ile Ile Ser Asn Glu Lys Gln Glu Ile Leu
1355 1360 1365
Gly Thr Val Ser Trp Asn Leu Arg Glu Met Leu Ala His Ala Glu
1370 1375 1380
Glu Thr Arg Lys Leu Met Pro Val Cys Val Glu Thr Lys Ala Ile
1385 1390 1395
Val Ser Thr Ile Gln Arg Lys Tyr Lys Gly Ile Lys Ile Gln Glu
1400 1405 1410
Gly Val Val Asp Tyr Gly Ala Arg Phe Tyr Phe Tyr Thr Ser Lys
1415 1420 1425
Thr Thr Val Ala Ser Leu Ile Asn Thr Leu Asn Asp Leu Asn Glu
1430 1435 1440
Thr Leu Val Thr Met Pro Leu Gly Tyr Val Thr His Gly Leu Asn
1445 1450 1455
Leu Glu Glu Ala Ala Arg Tyr Met Arg Ser Leu Lys Val Pro Ala
1460 1465 1470
Thr Val Ser Val Ser Ser Pro Asp Ala Val Thr Ala Tyr Asn Gly
1475 1480 1485
Tyr Leu Thr Ser Ser Ser Lys Thr Pro Glu Glu His Phe Ile Glu
1490 1495 1500
Thr Ile Ser Leu Ala Gly Ser Tyr Lys Asp Trp Ser Tyr Ser Gly
1505 1510 1515
Gln Ser Thr Gln Leu Gly Ile Glu Phe Leu Lys Arg Gly Asp Lys
1520 1525 1530
Ser Val Tyr Tyr Thr Ser Asn Pro Thr Thr Phe His Leu Asp Gly
1535 1540 1545
Glu Val Ile Thr Phe Asp Asn Leu Lys Thr Leu Leu Ser Leu Arg
1550 1555 1560
Glu Val Arg Thr Ile Lys Val Phe Thr Thr Val Asp Asn Ile Asn
1565 1570 1575
Leu His Thr Gln Val Val Asp Met Ser Met Thr Tyr Gly Gln Gln
1580 1585 1590
Phe Gly Pro Thr Tyr Leu Asp Gly Ala Asp Val Thr Lys Ile Lys
1595 1600 1605
Pro His Asn Ser His Glu Gly Lys Thr Phe Tyr Val Leu Pro Asn
1610 1615 1620
Asp Asp Thr Leu Arg Val Glu Ala Phe Glu Tyr Tyr His Thr Thr
1625 1630 1635
Asp Pro Ser Phe Leu Gly Arg Tyr Met Ser Ala Leu Asn His Thr
1640 1645 1650
Lys Lys Trp Lys Tyr Pro Gln Val Asn Gly Leu Thr Ser Ile Lys
1655 1660 1665
Trp Ala Asp Asn Asn Cys Tyr Leu Ala Thr Ala Leu Leu Thr Leu
1670 1675 1680
Gln Gln Ile Glu Leu Lys Phe Asn Pro Pro Ala Leu Gln Asp Ala
1685 1690 1695
Tyr Tyr Arg Ala Arg Ala Gly Glu Ala Ala Asn Phe Cys Ala Leu
1700 1705 1710
Ile Leu Ala Tyr Cys Asn Lys Thr Val Gly Glu Leu Gly Asp Val
1715 1720 1725
Arg Glu Thr Met Ser Tyr Leu Phe Gln His Ala Asn Leu Asp Ser
1730 1735 1740
Cys Lys Arg Val Leu Asn Val Val Cys Lys Thr Cys Gly Gln Gln
1745 1750 1755
Gln Thr Thr Leu Lys Gly Val Glu Ala Val Met Tyr Met Gly Thr
1760 1765 1770
Leu Ser Tyr Glu Gln Phe Lys Lys Gly Val Gln Ile Pro Cys Thr
1775 1780 1785
Cys Gly Lys Gln Ala Thr Lys Tyr Leu Val Gln Gln Glu Ser Pro
1790 1795 1800
Phe Val Met Met Ser Ala Pro Pro Ala Gln Tyr Glu Leu Lys His
1805 1810 1815
Gly Thr Phe Thr Cys Ala Ser Glu Tyr Thr Gly Asn Tyr Gln Cys
1820 1825 1830
Gly His Tyr Lys His Ile Thr Ser Lys Glu Thr Leu Tyr Cys Ile
1835 1840 1845
Asp Gly Ala Leu Leu Thr Lys Ser Ser Glu Tyr Lys Gly Pro Ile
1850 1855 1860
Thr Asp Val Phe Tyr Lys Glu Asn Ser Tyr Thr Thr Thr Ile Lys
1865 1870 1875
Pro Val Thr Tyr Lys Leu Asp Gly Val Val Cys Thr Glu Ile Asp
1880 1885 1890
Pro Lys Leu Asp Asn Tyr Tyr Lys Lys Asp Asn Ser Tyr Phe Thr
1895 1900 1905
Glu Gln Pro Ile Asp Leu Val Pro Asn Gln Pro Tyr Pro Asn Ala
1910 1915 1920
Ser Phe Asp Asn Phe Lys Phe Val Cys Asp Asn Ile Lys Phe Ala
1925 1930 1935
Asp Asp Leu Asn Gln Leu Thr Gly Tyr Lys Lys Pro Ala Ser Arg
1940 1945 1950
Glu Leu Lys Val Thr Phe Phe Pro Asp Leu Asn Gly Asp Val Val
1955 1960 1965
Ala Ile Asp Tyr Lys His Tyr Thr Pro Ser Phe Lys Lys Gly Ala
1970 1975 1980
Lys Leu Leu His Lys Pro Ile Val Trp His Val Asn Asn Ala Thr
1985 1990 1995
Asn Lys Ala Thr Tyr Lys Pro Asn Thr Trp Cys Ile Arg Cys Leu
2000 2005 2010
Trp Ser Thr Lys Pro Val Glu Thr Ser Asn Ser Phe Asp Val Leu
2015 2020 2025
Lys Ser Glu Asp Ala Gln Gly Met Asp Asn Leu Ala Cys Glu Asp
2030 2035 2040
Leu Lys Pro Val Ser Glu Glu Val Val Glu Asn Pro Thr Ile Gln
2045 2050 2055
Lys Asp Val Leu Glu Cys Asn Val Lys Thr Thr Glu Val Val Gly
2060 2065 2070
Asp Ile Ile Leu Lys Pro Ala Asn Asn Ser Leu Lys Ile Thr Glu
2075 2080 2085
Glu Val Gly His Thr Asp Leu Met Ala Ala Tyr Val Asp Asn Ser
2090 2095 2100
Ser Leu Thr Ile Lys Lys Pro Asn Glu Leu Ser Arg Val Leu Gly
2105 2110 2115
Leu Lys Thr Leu Ala Thr His Gly Leu Ala Ala Val Asn Ser Val
2120 2125 2130
Pro Trp Asp Thr Ile Ala Asn Tyr Ala Lys Pro Phe Leu Asn Lys
2135 2140 2145
Val Val Ser Thr Thr Thr Asn Ile Val Thr Arg Cys Leu Asn Arg
2150 2155 2160
Val Cys Thr Asn Tyr Met Pro Tyr Phe Phe Thr Leu Leu Leu Gln
2165 2170 2175
Leu Cys Thr Phe Thr Arg Ser Thr Asn Ser Arg Ile Lys Ala Ser
2180 2185 2190
Met Pro Thr Thr Ile Ala Lys Asn Thr Val Lys Ser Val Gly Lys
2195 2200 2205
Phe Cys Leu Glu Ala Ser Phe Asn Tyr Leu Lys Ser Pro Asn Phe
2210 2215 2220
Ser Lys Leu Ile Asn Ile Ile Ile Trp Phe Leu Leu Leu Ser Val
2225 2230 2235
Cys Leu Gly Ser Leu Ile Tyr Ser Thr Ala Ala Leu Gly Val Leu
2240 2245 2250
Met Ser Asn Leu Gly Met Pro Ser Tyr Cys Thr Gly Tyr Arg Glu
2255 2260 2265
Gly Tyr Leu Asn Ser Thr Asn Val Thr Ile Ala Thr Tyr Cys Thr
2270 2275 2280
Gly Ser Ile Pro Cys Ser Val Cys Leu Ser Gly Leu Asp Ser Leu
2285 2290 2295
Asp Thr Tyr Pro Ser Leu Glu Thr Ile Gln Ile Thr Ile Ser Ser
2300 2305 2310
Phe Lys Trp Asp Leu Thr Ala Phe Gly Leu Val Ala Glu Trp Phe
2315 2320 2325
Leu Ala Tyr Ile Leu Phe Thr Arg Phe Phe Tyr Val Leu Gly Leu
2330 2335 2340
Ala Ala Ile Met Gln Leu Phe Phe Ser Tyr Phe Ala Val His Phe
2345 2350 2355
Ile Ser Asn Ser Trp Leu Met Trp Leu Ile Ile Asn Leu Val Gln
2360 2365 2370
Met Ala Pro Ile Ser Ala Met Val Arg Met Tyr Ile Phe Phe Ala
2375 2380 2385
Ser Phe Tyr Tyr Val Trp Lys Ser Tyr Val His Val Val Asp Gly
2390 2395 2400
Cys Asn Ser Ser Thr Cys Met Met Cys Tyr Lys Arg Asn Arg Ala
2405 2410 2415
Thr Arg Val Glu Cys Thr Thr Ile Val Asn Gly Val Arg Arg Ser
2420 2425 2430
Phe Tyr Val Tyr Ala Asn Gly Gly Lys Gly Phe Cys Lys Leu His
2435 2440 2445
Asn Trp Asn Cys Val Asn Cys Asp Thr Phe Cys Ala Gly Ser Thr
2450 2455 2460
Phe Ile Ser Asp Glu Val Ala Arg Asp Leu Ser Leu Gln Phe Lys
2465 2470 2475
Arg Pro Ile Asn Pro Thr Asp Gln Ser Ser Tyr Ile Val Asp Ser
2480 2485 2490
Val Thr Val Lys Asn Gly Ser Ile His Leu Tyr Phe Asp Lys Ala
2495 2500 2505
Gly Gln Lys Thr Tyr Glu Arg His Ser Leu Ser His Phe Val Asn
2510 2515 2520
Leu Asp Asn Leu Arg Ala Asn Asn Thr Lys Gly Ser Leu Pro Ile
2525 2530 2535
Asn Val Ile Val Phe Asp Gly Lys Ser Lys Cys Glu Glu Ser Ser
2540 2545 2550
Ala Lys Ser Ala Ser Val Tyr Tyr Ser Gln Leu Met Cys Gln Pro
2555 2560 2565
Ile Leu Leu Leu Asp Gln Ala Leu Val Ser Asp Val Gly Asp Ser
2570 2575 2580
Ala Glu Val Ala Val Lys Met Phe Asp Ala Tyr Val Asn Thr Phe
2585 2590 2595
Ser Ser Thr Phe Asn Val Pro Met Glu Lys Leu Lys Thr Leu Val
2600 2605 2610
Ala Thr Ala Glu Ala Glu Leu Ala Lys Asn Val Ser Leu Asp Asn
2615 2620 2625
Val Leu Ser Thr Phe Ile Ser Ala Ala Arg Gln Gly Phe Val Asp
2630 2635 2640
Ser Asp Val Glu Thr Lys Asp Val Val Glu Cys Leu Lys Leu Ser
2645 2650 2655
His Gln Ser Asp Ile Glu Val Thr Gly Asp Ser Cys Asn Asn Tyr
2660 2665 2670
Met Leu Thr Tyr Asn Lys Val Glu Asn Met Thr Pro Arg Asp Leu
2675 2680 2685
Gly Ala Cys Ile Asp Cys Ser Ala Arg His Ile Asn Ala Gln Val
2690 2695 2700
Ala Lys Ser His Asn Ile Ala Leu Ile Trp Asn Val Lys Asp Phe
2705 2710 2715
Met Ser Leu Ser Glu Gln Leu Arg Lys Gln Ile Arg Ser Ala Ala
2720 2725 2730
Lys Lys Asn Asn Leu Pro Phe Lys Leu Thr Cys Ala Thr Thr Arg
2735 2740 2745
Gln Val Val Asn Val Val Thr Thr Lys Ile Ala Leu Lys Gly Gly
2750 2755 2760
Lys Ile Val Asn Asn Trp Leu Lys Gln Leu Ile Lys Val Thr Leu
2765 2770 2775
Val Phe Leu Phe Val Ala Ala Ile Phe Tyr Leu Ile Thr Pro Val
2780 2785 2790
His Val Met Ser Lys His Thr Asp Phe Ser Ser Glu Ile Ile Gly
2795 2800 2805
Tyr Lys Ala Ile Asp Gly Gly Val Thr Arg Asp Ile Ala Ser Thr
2810 2815 2820
Asp Thr Cys Phe Ala Asn Lys His Ala Asp Phe Asp Thr Trp Phe
2825 2830 2835
Ser Gln Arg Gly Gly Ser Tyr Thr Asn Asp Lys Ala Cys Pro Leu
2840 2845 2850
Ile Ala Ala Val Ile Thr Arg Glu Val Gly Phe Val Val Pro Gly
2855 2860 2865
Leu Pro Gly Thr Ile Leu Arg Thr Thr Asn Gly Asp Phe Leu His
2870 2875 2880
Phe Leu Pro Arg Val Phe Ser Ala Val Gly Asn Ile Cys Tyr Thr
2885 2890 2895
Pro Ser Lys Leu Ile Glu Tyr Thr Asp Phe Ala Thr Ser Ala Cys
2900 2905 2910
Val Leu Ala Ala Glu Cys Thr Ile Phe Lys Asp Ala Ser Gly Lys
2915 2920 2925
Pro Val Pro Tyr Cys Tyr Asp Thr Asn Val Leu Glu Gly Ser Val
2930 2935 2940
Ala Tyr Glu Ser Leu Arg Pro Asp Thr Arg Tyr Val Leu Met Asp
2945 2950 2955
Gly Ser Ile Ile Gln Phe Pro Asn Thr Tyr Leu Glu Gly Ser Val
2960 2965 2970
Arg Val Val Thr Thr Phe Asp Ser Glu Tyr Cys Arg His Gly Thr
2975 2980 2985
Cys Glu Arg Ser Glu Ala Gly Val Cys Val Ser Thr Ser Gly Arg
2990 2995 3000
Trp Val Leu Asn Asn Asp Tyr Tyr Arg Ser Leu Pro Gly Val Phe
3005 3010 3015
Cys Gly Val Asp Ala Val Asn Leu Leu Thr Asn Met Phe Thr Pro
3020 3025 3030
Leu Ile Gln Pro Ile Gly Ala Leu Asp Ile Ser Ala Ser Ile Val
3035 3040 3045
Ala Gly Gly Ile Val Ala Ile Val Val Thr Cys Leu Ala Tyr Tyr
3050 3055 3060
Phe Met Arg Phe Arg Arg Ala Phe Gly Glu Tyr Ser His Val Val
3065 3070 3075
Ala Phe Asn Thr Leu Leu Phe Leu Met Ser Phe Thr Val Leu Cys
3080 3085 3090
Leu Thr Pro Val Tyr Ser Phe Leu Pro Gly Val Tyr Ser Val Ile
3095 3100 3105
Tyr Leu Tyr Leu Thr Phe Tyr Leu Thr Asn Asp Val Ser Phe Leu
3110 3115 3120
Ala His Ile Gln Trp Met Val Met Phe Thr Pro Leu Val Pro Phe
3125 3130 3135
Trp Ile Thr Ile Ala Tyr Ile Ile Cys Ile Ser Thr Lys His Phe
3140 3145 3150
Tyr Trp Phe Phe Ser Asn Tyr Leu Lys Arg Arg Val Val Phe Asn
3155 3160 3165
Gly Val Ser Phe Ser Thr Phe Glu Glu Ala Ala Leu Cys Thr Phe
3170 3175 3180
Leu Leu Asn Lys Glu Met Tyr Leu Lys Leu Arg Ser Asp Val Leu
3185 3190 3195
Leu Pro Leu Thr Gln Tyr Asn Arg Tyr Leu Ala Leu Tyr Asn Lys
3200 3205 3210
Tyr Lys Tyr Phe Ser Gly Ala Met Asp Thr Thr Ser Tyr Arg Glu
3215 3220 3225
Ala Ala Cys Cys His Leu Ala Lys Ala Leu Asn Asp Phe Ser Asn
3230 3235 3240
Ser Gly Ser Asp Val Leu Tyr Gln Pro Pro Gln Thr Ser Ile Thr
3245 3250 3255
Ser Ala Val Leu Gln Ser Gly Phe Arg Lys Met Ala Phe Pro Ser
3260 3265 3270
Gly Lys Val Glu Gly Cys Met Val Gln Val Thr Cys Gly Thr Thr
3275 3280 3285
Thr Leu Asn Gly Leu Trp Leu Asp Asp Val Val Tyr Cys Pro Arg
3290 3295 3300
His Val Ile Cys Thr Ser Glu Asp Met Leu Asn Pro Asn Tyr Glu
3305 3310 3315
Asp Leu Leu Ile Arg Lys Ser Asn His Asn Phe Leu Val Gln Ala
3320 3325 3330
Gly Asn Val Gln Leu Arg Val Ile Gly His Ser Met Gln Asn Cys
3335 3340 3345
Val Leu Lys Leu Lys Val Asp Thr Ala Asn Pro Lys Thr Pro Lys
3350 3355 3360
Tyr Lys Phe Val Arg Ile Gln Pro Gly Gln Thr Phe Ser Val Leu
3365 3370 3375
Ala Cys Tyr Asn Gly Ser Pro Ser Gly Val Tyr Gln Cys Ala Met
3380 3385 3390
Arg Pro Asn Phe Thr Ile Lys Gly Ser Phe Leu Asn Gly Ser Cys
3395 3400 3405
Gly Ser Val Gly Phe Asn Ile Asp Tyr Asp Cys Val Ser Phe Cys
3410 3415 3420
Tyr Met His His Met Glu Leu Pro Thr Gly Val His Ala Gly Thr
3425 3430 3435
Asp Leu Glu Gly Asn Phe Tyr Gly Pro Phe Val Asp Arg Gln Thr
3440 3445 3450
Ala Gln Ala Ala Gly Thr Asp Thr Thr Ile Thr Val Asn Val Leu
3455 3460 3465
Ala Trp Leu Tyr Ala Ala Val Ile Asn Gly Asp Arg Trp Phe Leu
3470 3475 3480
Asn Arg Phe Thr Thr Thr Leu Asn Asp Phe Asn Leu Val Ala Met
3485 3490 3495
Lys Tyr Asn Tyr Glu Pro Leu Thr Gln Asp His Val Asp Ile Leu
3500 3505 3510
Gly Pro Leu Ser Ala Gln Thr Gly Ile Ala Val Leu Asp Met Cys
3515 3520 3525
Ala Ser Leu Lys Glu Leu Leu Gln Asn Gly Met Asn Gly Arg Thr
3530 3535 3540
Ile Leu Gly Ser Ala Leu Leu Glu Asp Glu Phe Thr Pro Phe Asp
3545 3550 3555
Val Val Arg Gln Cys Ser Gly Val Thr Phe Gln Ser Ala Val Lys
3560 3565 3570
Arg Thr Ile Lys Gly Thr His His Trp Leu Leu Leu Thr Ile Leu
3575 3580 3585
Thr Ser Leu Leu Val Leu Val Gln Ser Thr Gln Trp Ser Leu Phe
3590 3595 3600
Phe Phe Leu Tyr Glu Asn Ala Phe Leu Pro Phe Ala Met Gly Ile
3605 3610 3615
Ile Ala Met Ser Ala Phe Ala Met Met Phe Val Lys His Lys His
3620 3625 3630
Ala Phe Leu Cys Leu Phe Leu Leu Pro Ser Leu Ala Thr Val Ala
3635 3640 3645
Tyr Phe Asn Met Val Tyr Met Pro Ala Ser Trp Val Met Arg Ile
3650 3655 3660
Met Thr Trp Leu Asp Met Val Asp Thr Ser Leu Ser Gly Phe Lys
3665 3670 3675
Leu Lys Asp Cys Val Met Tyr Ala Ser Ala Val Val Leu Leu Ile
3680 3685 3690
Leu Met Thr Ala Arg Thr Val Tyr Asp Asp Gly Ala Arg Arg Val
3695 3700 3705
Trp Thr Leu Met Asn Val Leu Thr Leu Val Tyr Lys Val Tyr Tyr
3710 3715 3720
Gly Asn Ala Leu Asp Gln Ala Ile Ser Met Trp Ala Leu Ile Ile
3725 3730 3735
Ser Val Thr Ser Asn Tyr Ser Gly Val Val Thr Thr Val Met Phe
3740 3745 3750
Leu Ala Arg Gly Ile Val Phe Met Cys Val Glu Tyr Cys Pro Ile
3755 3760 3765
Phe Phe Ile Thr Gly Asn Thr Leu Gln Cys Ile Met Leu Val Tyr
3770 3775 3780
Cys Phe Leu Gly Tyr Phe Cys Thr Cys Tyr Phe Gly Leu Phe Cys
3785 3790 3795
Leu Leu Asn Arg Tyr Phe Arg Leu Thr Leu Gly Val Tyr Asp Tyr
3800 3805 3810
Leu Val Ser Thr Gln Glu Phe Arg Tyr Met Asn Ser Gln Gly Leu
3815 3820 3825
Leu Pro Pro Lys Asn Ser Ile Asp Ala Phe Lys Leu Asn Ile Lys
3830 3835 3840
Leu Leu Gly Val Gly Gly Lys Pro Cys Ile Lys Val Ala Thr Val
3845 3850 3855
Gln Ser Lys Met Ser Asp Val Lys Cys Thr Ser Val Val Leu Leu
3860 3865 3870
Ser Val Leu Gln Gln Leu Arg Val Glu Ser Ser Ser Lys Leu Trp
3875 3880 3885
Ala Gln Cys Val Gln Leu His Asn Asp Ile Leu Leu Ala Lys Asp
3890 3895 3900
Thr Thr Glu Ala Phe Glu Lys Met Val Ser Leu Leu Ser Val Leu
3905 3910 3915
Leu Ser Met Gln Gly Ala Val Asp Ile Asn Lys Leu Cys Glu Glu
3920 3925 3930
Met Leu Asp Asn Arg Ala Thr Leu Gln Ala Ile Ala Ser Glu Phe
3935 3940 3945
Ser Ser Leu Pro Ser Tyr Ala Ala Phe Ala Thr Ala Gln Glu Ala
3950 3955 3960
Tyr Glu Gln Ala Val Ala Asn Gly Asp Ser Glu Val Val Leu Lys
3965 3970 3975
Lys Leu Lys Lys Ser Leu Asn Val Ala Lys Ser Glu Phe Asp Arg
3980 3985 3990
Asp Ala Ala Met Gln Arg Lys Leu Glu Lys Met Ala Asp Gln Ala
3995 4000 4005
Met Thr Gln Met Tyr Lys Gln Ala Arg Ser Glu Asp Lys Arg Ala
4010 4015 4020
Lys Val Thr Ser Ala Met Gln Thr Met Leu Phe Thr Met Leu Arg
4025 4030 4035
Lys Leu Asp Asn Asp Ala Leu Asn Asn Ile Ile Asn Asn Ala Arg
4040 4045 4050
Asp Gly Cys Val Pro Leu Asn Ile Ile Pro Leu Thr Thr Ala Ala
4055 4060 4065
Lys Leu Met Val Val Ile Pro Asp Tyr Asn Thr Tyr Lys Asn Thr
4070 4075 4080
Cys Asp Gly Thr Thr Phe Thr Tyr Ala Ser Ala Leu Trp Glu Ile
4085 4090 4095
Gln Gln Val Val Asp Ala Asp Ser Lys Ile Val Gln Leu Ser Glu
4100 4105 4110
Ile Ser Met Asp Asn Ser Pro Asn Leu Ala Trp Pro Leu Ile Val
4115 4120 4125
Thr Ala Leu Arg Ala Asn Ser Ala Val Lys Leu Gln Asn Asn Glu
4130 4135 4140
Leu Ser Pro Val Ala Leu Arg Gln Met Ser Cys Ala Ala Gly Thr
4145 4150 4155
Thr Gln Thr Ala Cys Thr Asp Asp Asn Ala Leu Ala Tyr Tyr Asn
4160 4165 4170
Thr Thr Lys Gly Gly Arg Phe Val Leu Ala Leu Leu Ser Asp Leu
4175 4180 4185
Gln Asp Leu Lys Trp Ala Arg Phe Pro Lys Ser Asp Gly Thr Gly
4190 4195 4200
Thr Ile Tyr Thr Glu Leu Glu Pro Pro Cys Arg Phe Val Thr Asp
4205 4210 4215
Thr Pro Lys Gly Pro Lys Val Lys Tyr Leu Tyr Phe Ile Lys Gly
4220 4225 4230
Leu Asn Asn Leu Asn Arg Gly Met Val Leu Gly Ser Leu Ala Ala
4235 4240 4245
Thr Val Arg Leu Gln Ala Gly Asn Ala Thr Glu Val Pro Ala Asn
4250 4255 4260
Ser Thr Val Leu Ser Phe Cys Ala Phe Ala Val Asp Ala Ala Lys
4265 4270 4275
Ala Tyr Lys Asp Tyr Leu Ala Ser Gly Gly Gln Pro Ile Thr Asn
4280 4285 4290
Cys Val Lys Met Leu Cys Thr His Thr Gly Thr Gly Gln Ala Ile
4295 4300 4305
Thr Val Thr Pro Glu Ala Asn Met Asp Gln Glu Ser Phe Gly Gly
4310 4315 4320
Ala Ser Cys Cys Leu Tyr Cys Arg Cys His Ile Asp His Pro Asn
4325 4330 4335
Pro Lys Gly Phe Cys Asp Leu Lys Gly Lys Tyr Val Gln Ile Pro
4340 4345 4350
Thr Thr Cys Ala Asn Asp Pro Val Gly Phe Thr Leu Lys Asn Thr
4355 4360 4365
Val Cys Thr Val Cys Gly Met Trp Lys Gly Tyr Gly Cys Ser Cys
4370 4375 4380
Asp Gln Leu Arg Glu Pro Met Leu Gln Ser Ala Asp Ala Gln Ser
4385 4390 4395
Phe Leu Asn Arg Val Cys Gly Val Ser Ala Ala Arg Leu Thr Pro
4400 4405 4410
Cys Gly Thr Gly Thr Ser Thr Asp Val Val Tyr Arg Ala Phe Asp
4415 4420 4425
Ile Tyr Asn Asp Lys Val Ala Gly Phe Ala Lys Phe Leu Lys Thr
4430 4435 4440
Asn Cys Cys Arg Phe Gln Glu Lys Asp Glu Asp Asp Asn Leu Ile
4445 4450 4455
Asp Ser Tyr Phe Val Val Lys Arg His Thr Phe Ser Asn Tyr Gln
4460 4465 4470
His Glu Glu Thr Ile Tyr Asn Leu Leu Lys Asp Cys Pro Ala Val
4475 4480 4485
Ala Lys His Asp Phe Phe Lys Phe Arg Ile Asp Gly Asp Met Val
4490 4495 4500
Pro His Ile Ser Arg Gln Arg Leu Thr Lys Tyr Thr Met Ala Asp
4505 4510 4515
Leu Val Tyr Ala Leu Arg His Phe Asp Glu Gly Asn Cys Asp Thr
4520 4525 4530
Leu Lys Glu Ile Leu Val Thr Tyr Asn Cys Cys Asp Asp Asp Tyr
4535 4540 4545
Phe Asn Lys Lys Asp Trp Tyr Asp Phe Val Glu Asn Pro Asp Ile
4550 4555 4560
Leu Arg Val Tyr Ala Asn Leu Gly Glu Arg Val Arg Gln Ala Leu
4565 4570 4575
Leu Lys Thr Val Gln Phe Cys Asp Ala Met Arg Asn Ala Gly Ile
4580 4585 4590
Val Gly Val Leu Thr Leu Asp Asn Gln Asp Leu Asn Gly Asn Trp
4595 4600 4605
Tyr Asp Phe Gly Asp Phe Ile Gln Thr Thr Pro Gly Ser Gly Val
4610 4615 4620
Pro Val Val Asp Ser Tyr Tyr Ser Leu Leu Met Pro Ile Leu Thr
4625 4630 4635
Leu Thr Arg Ala Leu Thr Ala Glu Ser His Val Asp Thr Asp Leu
4640 4645 4650
Thr Lys Pro Tyr Ile Lys Trp Asp Leu Leu Lys Tyr Asp Phe Thr
4655 4660 4665
Glu Glu Arg Leu Lys Leu Phe Asp Arg Tyr Phe Lys Tyr Trp Asp
4670 4675 4680
Gln Thr Tyr His Pro Asn Cys Val Asn Cys Leu Asp Asp Arg Cys
4685 4690 4695
Ile Leu His Cys Ala Asn Phe Asn Val Leu Phe Ser Thr Val Phe
4700 4705 4710
Pro Pro Thr Ser Phe Gly Pro Leu Val Arg Lys Ile Phe Val Asp
4715 4720 4725
Gly Val Pro Phe Val Val Ser Thr Gly Tyr His Phe Arg Glu Leu
4730 4735 4740
Gly Val Val His Asn Gln Asp Val Asn Leu His Ser Ser Arg Leu
4745 4750 4755
Ser Phe Lys Glu Leu Leu Val Tyr Ala Ala Asp Pro Ala Met His
4760 4765 4770
Ala Ala Ser Gly Asn Leu Leu Leu Asp Lys Arg Thr Thr Cys Phe
4775 4780 4785
Ser Val Ala Ala Leu Thr Asn Asn Val Ala Phe Gln Thr Val Lys
4790 4795 4800
Pro Gly Asn Phe Asn Lys Asp Phe Tyr Asp Phe Ala Val Ser Lys
4805 4810 4815
Gly Phe Phe Lys Glu Gly Ser Ser Val Glu Leu Lys His Phe Phe
4820 4825 4830
Phe Ala Gln Asp Gly Asn Ala Ala Ile Ser Asp Tyr Asp Tyr Tyr
4835 4840 4845
Arg Tyr Asn Leu Pro Thr Met Cys Asp Ile Arg Gln Leu Leu Phe
4850 4855 4860
Val Val Glu Val Val Asp Lys Tyr Phe Asp Cys Tyr Asp Gly Gly
4865 4870 4875
Cys Ile Asn Ala Asn Gln Val Ile Val Asn Asn Leu Asp Lys Ser
4880 4885 4890
Ala Gly Phe Pro Phe Asn Lys Trp Gly Lys Ala Arg Leu Tyr Tyr
4895 4900 4905
Asp Ser Met Ser Tyr Glu Asp Gln Asp Ala Leu Phe Ala Tyr Thr
4910 4915 4920
Lys Arg Asn Val Ile Pro Thr Ile Thr Gln Met Asn Leu Lys Tyr
4925 4930 4935
Ala Ile Ser Ala Lys Asn Arg Ala Arg Thr Val Ala Gly Val Ser
4940 4945 4950
Ile Cys Ser Thr Met Thr Asn Arg Gln Phe His Gln Lys Leu Leu
4955 4960 4965
Lys Ser Ile Ala Ala Thr Arg Gly Ala Thr Val Val Ile Gly Thr
4970 4975 4980
Ser Lys Phe Tyr Gly Gly Trp His Asn Met Leu Lys Thr Val Tyr
4985 4990 4995
Ser Asp Val Glu Asn Pro His Leu Met Gly Trp Asp Tyr Pro Lys
5000 5005 5010
Cys Asp Arg Ala Met Pro Asn Met Leu Arg Ile Met Ala Ser Leu
5015 5020 5025
Val Leu Ala Arg Lys His Thr Thr Cys Cys Ser Leu Ser His Arg
5030 5035 5040
Phe Tyr Arg Leu Ala Asn Glu Cys Ala Gln Val Leu Ser Glu Met
5045 5050 5055
Val Met Cys Gly Gly Ser Leu Tyr Val Lys Pro Gly Gly Thr Ser
5060 5065 5070
Ser Gly Asp Ala Thr Thr Ala Tyr Ala Asn Ser Val Phe Asn Ile
5075 5080 5085
Cys Gln Ala Val Thr Ala Asn Val Asn Ala Leu Leu Ser Thr Asp
5090 5095 5100
Gly Asn Lys Ile Ala Asp Lys Tyr Val Arg Asn Leu Gln His Arg
5105 5110 5115
Leu Tyr Glu Cys Leu Tyr Arg Asn Arg Asp Val Asp Thr Asp Phe
5120 5125 5130
Val Asn Glu Phe Tyr Ala Tyr Leu Arg Lys His Phe Ser Met Met
5135 5140 5145
Ile Leu Ser Asp Asp Ala Val Val Cys Phe Asn Ser Thr Tyr Ala
5150 5155 5160
Ser Gln Gly Leu Val Ala Ser Ile Lys Asn Phe Lys Ser Val Leu
5165 5170 5175
Tyr Tyr Gln Asn Asn Val Phe Met Ser Glu Ala Lys Cys Trp Thr
5180 5185 5190
Glu Thr Asp Leu Thr Lys Gly Pro His Glu Phe Cys Ser Gln His
5195 5200 5205
Thr Met Leu Val Lys Gln Gly Asp Asp Tyr Val Tyr Leu Pro Tyr
5210 5215 5220
Pro Asp Pro Ser Arg Ile Leu Gly Ala Gly Cys Phe Val Asp Asp
5225 5230 5235
Ile Val Lys Thr Asp Gly Thr Leu Met Ile Glu Arg Phe Val Ser
5240 5245 5250
Leu Ala Ile Asp Ala Tyr Pro Leu Thr Lys His Pro Asn Gln Glu
5255 5260 5265
Tyr Ala Asp Val Phe His Leu Tyr Leu Gln Tyr Ile Arg Lys Leu
5270 5275 5280
His Asp Glu Leu Thr Gly His Met Leu Asp Met Tyr Ser Val Met
5285 5290 5295
Leu Thr Asn Asp Asn Thr Ser Arg Tyr Trp Glu Pro Glu Phe Tyr
5300 5305 5310
Glu Ala Met Tyr Thr Pro His Thr Val Leu Gln Ala Val Gly Ala
5315 5320 5325
Cys Val Leu Cys Asn Ser Gln Thr Ser Leu Arg Cys Gly Ala Cys
5330 5335 5340
Ile Arg Arg Pro Phe Leu Cys Cys Lys Cys Cys Tyr Asp His Val
5345 5350 5355
Ile Ser Thr Ser His Lys Leu Val Leu Ser Val Asn Pro Tyr Val
5360 5365 5370
Cys Asn Ala Pro Gly Cys Asp Val Thr Asp Val Thr Gln Leu Tyr
5375 5380 5385
Leu Gly Gly Met Ser Tyr Tyr Cys Lys Ser His Lys Pro Pro Ile
5390 5395 5400
Ser Phe Pro Leu Cys Ala Asn Gly Gln Val Phe Gly Leu Tyr Lys
5405 5410 5415
Asn Thr Cys Val Gly Ser Asp Asn Val Thr Asp Phe Asn Ala Ile
5420 5425 5430
Ala Thr Cys Asp Trp Thr Asn Ala Gly Asp Tyr Ile Leu Ala Asn
5435 5440 5445
Thr Cys Thr Glu Arg Leu Lys Leu Phe Ala Ala Glu Thr Leu Lys
5450 5455 5460
Ala Thr Glu Glu Thr Phe Lys Leu Ser Tyr Gly Ile Ala Thr Val
5465 5470 5475
Arg Glu Val Leu Ser Asp Arg Glu Leu His Leu Ser Trp Glu Val
5480 5485 5490
Gly Lys Pro Arg Pro Pro Leu Asn Arg Asn Tyr Val Phe Thr Gly
5495 5500 5505
Tyr Arg Val Thr Lys Asn Ser Lys Val Gln Ile Gly Glu Tyr Thr
5510 5515 5520
Phe Glu Lys Gly Asp Tyr Gly Asp Ala Val Val Tyr Arg Gly Thr
5525 5530 5535
Thr Thr Tyr Lys Leu Asn Val Gly Asp Tyr Phe Val Leu Thr Ser
5540 5545 5550
His Thr Val Met Pro Leu Ser Ala Pro Thr Leu Val Pro Gln Glu
5555 5560 5565
His Tyr Val Arg Ile Thr Gly Leu Tyr Pro Thr Leu Asn Ile Ser
5570 5575 5580
Asp Glu Phe Ser Ser Asn Val Ala Asn Tyr Gln Lys Val Gly Met
5585 5590 5595
Gln Lys Tyr Ser Thr Leu Gln Gly Pro Pro Gly Thr Gly Lys Ser
5600 5605 5610
His Phe Ala Ile Gly Leu Ala Leu Tyr Tyr Pro Ser Ala Arg Ile
5615 5620 5625
Val Tyr Thr Ala Cys Ser His Ala Ala Val Asp Ala Leu Cys Glu
5630 5635 5640
Lys Ala Leu Lys Tyr Leu Pro Ile Asp Lys Cys Ser Arg Ile Ile
5645 5650 5655
Pro Ala Arg Ala Arg Val Glu Cys Phe Asp Lys Phe Lys Val Asn
5660 5665 5670
Ser Thr Leu Glu Gln Tyr Val Phe Cys Thr Val Asn Ala Leu Pro
5675 5680 5685
Glu Thr Thr Ala Asp Ile Val Val Phe Asp Glu Ile Ser Met Ala
5690 5695 5700
Thr Asn Tyr Asp Leu Ser Val Val Asn Ala Arg Leu Arg Ala Lys
5705 5710 5715
His Tyr Val Tyr Ile Gly Asp Pro Ala Gln Leu Pro Ala Pro Arg
5720 5725 5730
Thr Leu Leu Thr Lys Gly Thr Leu Glu Pro Glu Tyr Phe Asn Ser
5735 5740 5745
Val Cys Arg Leu Met Lys Thr Ile Gly Pro Asp Met Phe Leu Gly
5750 5755 5760
Thr Cys Arg Arg Cys Pro Ala Glu Ile Val Asp Thr Val Ser Ala
5765 5770 5775
Leu Val Tyr Asp Asn Lys Leu Lys Ala His Lys Asp Lys Ser Ala
5780 5785 5790
Gln Cys Phe Lys Met Phe Tyr Lys Gly Val Ile Thr His Asp Val
5795 5800 5805
Ser Ser Ala Ile Asn Arg Pro Gln Ile Gly Val Val Arg Glu Phe
5810 5815 5820
Leu Thr Arg Asn Pro Ala Trp Arg Lys Ala Val Phe Ile Ser Pro
5825 5830 5835
Tyr Asn Ser Gln Asn Ala Val Ala Ser Lys Ile Leu Gly Leu Pro
5840 5845 5850
Thr Gln Thr Val Asp Ser Ser Gln Gly Ser Glu Tyr Asp Tyr Val
5855 5860 5865
Ile Phe Thr Gln Thr Thr Glu Thr Ala His Ser Cys Asn Val Asn
5870 5875 5880
Arg Phe Asn Val Ala Ile Thr Arg Ala Lys Val Gly Ile Leu Cys
5885 5890 5895
Ile Met Ser Asp Arg Asp Leu Tyr Asp Lys Leu Gln Phe Thr Ser
5900 5905 5910
Leu Glu Ile Pro Arg Arg Asn Val Ala Thr Leu Gln Ala Glu Asn
5915 5920 5925
Val Thr Gly Leu Phe Lys Asp Cys Ser Lys Val Ile Thr Gly Leu
5930 5935 5940
His Pro Thr Gln Ala Pro Thr His Leu Ser Val Asp Thr Lys Phe
5945 5950 5955
Lys Thr Glu Gly Leu Cys Val Asp Ile Pro Gly Ile Pro Lys Asp
5960 5965 5970
Met Thr Tyr Arg Arg Leu Ile Ser Met Met Gly Phe Lys Met Asn
5975 5980 5985
Tyr Gln Val Asn Gly Tyr Pro Asn Met Phe Ile Thr Arg Glu Glu
5990 5995 6000
Ala Ile Arg His Val Arg Ala Trp Ile Gly Phe Asp Val Glu Gly
6005 6010 6015
Cys His Ala Thr Arg Glu Ala Val Gly Thr Asn Leu Pro Leu Gln
6020 6025 6030
Leu Gly Phe Ser Thr Gly Val Asn Leu Val Ala Val Pro Thr Gly
6035 6040 6045
Tyr Val Asp Thr Pro Asn Asn Thr Asp Phe Ser Arg Val Ser Ala
6050 6055 6060
Lys Pro Pro Pro Gly Asp Gln Phe Lys His Leu Ile Pro Leu Met
6065 6070 6075
Tyr Lys Gly Leu Pro Trp Asn Val Val Arg Ile Lys Ile Val Gln
6080 6085 6090
Met Leu Ser Asp Thr Leu Lys Asn Leu Ser Asp Arg Val Val Phe
6095 6100 6105
Val Leu Trp Ala His Gly Phe Glu Leu Thr Ser Met Lys Tyr Phe
6110 6115 6120
Val Lys Ile Gly Pro Glu Arg Thr Cys Cys Leu Cys Asp Arg Arg
6125 6130 6135
Ala Thr Cys Phe Ser Thr Ala Ser Asp Thr Tyr Ala Cys Trp His
6140 6145 6150
His Ser Ile Gly Phe Asp Tyr Val Tyr Asn Pro Phe Met Ile Asp
6155 6160 6165
Val Gln Gln Trp Gly Phe Thr Gly Asn Leu Gln Ser Asn His Asp
6170 6175 6180
Leu Tyr Cys Gln Val His Gly Asn Ala His Val Ala Ser Cys Asp
6185 6190 6195
Ala Ile Met Thr Arg Cys Leu Ala Val His Glu Cys Phe Val Lys
6200 6205 6210
Arg Val Asp Trp Thr Ile Glu Tyr Pro Ile Ile Gly Asp Glu Leu
6215 6220 6225
Lys Ile Asn Ala Ala Cys Arg Lys Val Gln His Met Val Val Lys
6230 6235 6240
Ala Ala Leu Leu Ala Asp Lys Phe Pro Val Leu His Asp Ile Gly
6245 6250 6255
Asn Pro Lys Ala Ile Lys Cys Val Pro Gln Ala Asp Val Glu Trp
6260 6265 6270
Lys Phe Tyr Asp Ala Gln Pro Cys Ser Asp Lys Ala Tyr Lys Ile
6275 6280 6285
Glu Glu Leu Phe Tyr Ser Tyr Ala Thr His Ser Asp Lys Phe Thr
6290 6295 6300
Asp Gly Val Cys Leu Phe Trp Asn Cys Asn Val Asp Arg Tyr Pro
6305 6310 6315
Ala Asn Ser Ile Val Cys Arg Phe Asp Thr Arg Val Leu Ser Asn
6320 6325 6330
Leu Asn Leu Pro Gly Cys Asp Gly Gly Ser Leu Tyr Val Asn Lys
6335 6340 6345
His Ala Phe His Thr Pro Ala Phe Asp Lys Ser Ala Phe Val Asn
6350 6355 6360
Leu Lys Gln Leu Pro Phe Phe Tyr Tyr Ser Asp Ser Pro Cys Glu
6365 6370 6375
Ser His Gly Lys Gln Val Val Ser Asp Ile Asp Tyr Val Pro Leu
6380 6385 6390
Lys Ser Ala Thr Cys Ile Thr Arg Cys Asn Leu Gly Gly Ala Val
6395 6400 6405
Cys Arg His His Ala Asn Glu Tyr Arg Leu Tyr Leu Asp Ala Tyr
6410 6415 6420
Asn Met Met Ile Ser Ala Gly Phe Ser Leu Trp Val Tyr Lys Gln
6425 6430 6435
Phe Asp Thr Tyr Asn Leu Trp Asn Thr Phe Thr Arg Leu Gln Ser
6440 6445 6450
Leu Glu Asn Val Ala Phe Asn Val Val Asn Lys Gly His Phe Asp
6455 6460 6465
Gly Gln Gln Gly Glu Val Pro Val Ser Ile Ile Asn Asn Thr Val
6470 6475 6480
Tyr Thr Lys Val Asp Gly Val Asp Val Glu Leu Phe Glu Asn Lys
6485 6490 6495
Thr Thr Leu Pro Val Asn Val Ala Phe Glu Leu Trp Ala Lys Arg
6500 6505 6510
Asn Ile Lys Pro Val Pro Glu Val Lys Ile Leu Asn Asn Leu Gly
6515 6520 6525
Val Asp Ile Ala Ala Asn Thr Val Ile Trp Asp Tyr Lys Arg Asp
6530 6535 6540
Ala Pro Ala His Ile Ser Thr Ile Gly Val Cys Ser Met Thr Asp
6545 6550 6555
Ile Ala Lys Lys Pro Thr Glu Thr Ile Cys Ala Pro Leu Thr Val
6560 6565 6570
Phe Phe Asp Gly Arg Val Asp Gly Gln Val Asp Leu Phe Arg Asn
6575 6580 6585
Ala Arg Asn Gly Val Leu Ile Thr Glu Gly Ser Val Lys Gly Leu
6590 6595 6600
Gln Pro Ser Val Gly Pro Lys Gln Ala Ser Leu Asn Gly Val Thr
6605 6610 6615
Leu Ile Gly Glu Ala Val Lys Thr Gln Phe Asn Tyr Tyr Lys Lys
6620 6625 6630
Val Asp Gly Val Val Gln Gln Leu Pro Glu Thr Tyr Phe Thr Gln
6635 6640 6645
Ser Arg Asn Leu Gln Glu Phe Lys Pro Arg Ser Gln Met Glu Ile
6650 6655 6660
Asp Phe Leu Glu Leu Ala Met Asp Glu Phe Ile Glu Arg Tyr Lys
6665 6670 6675
Leu Glu Gly Tyr Ala Phe Glu His Ile Val Tyr Gly Asp Phe Ser
6680 6685 6690
His Ser Gln Leu Gly Gly Leu His Leu Leu Ile Gly Leu Ala Lys
6695 6700 6705
Arg Phe Lys Glu Ser Pro Phe Glu Leu Glu Asp Phe Ile Pro Met
6710 6715 6720
Asp Ser Thr Val Lys Asn Tyr Phe Ile Thr Asp Ala Gln Thr Gly
6725 6730 6735
Ser Ser Lys Cys Val Cys Ser Val Ile Asp Leu Leu Leu Asp Asp
6740 6745 6750
Phe Val Glu Ile Ile Lys Ser Gln Asp Leu Ser Val Val Ser Lys
6755 6760 6765
Val Val Lys Val Thr Ile Asp Tyr Thr Glu Ile Ser Phe Met Leu
6770 6775 6780
Trp Cys Lys Asp Gly His Val Glu Thr Phe Tyr Pro Lys Leu Gln
6785 6790 6795
Ser Ser Gln Ala Trp Gln Pro Gly Val Ala Met Pro Asn Leu Tyr
6800 6805 6810
Lys Met Gln Arg Met Leu Leu Glu Lys Cys Asp Leu Gln Asn Tyr
6815 6820 6825
Gly Asp Ser Ala Thr Leu Pro Lys Gly Ile Met Met Asn Val Ala
6830 6835 6840
Lys Tyr Thr Gln Leu Cys Gln Tyr Leu Asn Thr Leu Thr Leu Ala
6845 6850 6855
Val Pro Tyr Asn Met Arg Val Ile His Phe Gly Ala Gly Ser Asp
6860 6865 6870
Lys Gly Val Ala Pro Gly Thr Ala Val Leu Arg Gln Trp Leu Pro
6875 6880 6885
Thr Gly Thr Leu Leu Val Asp Ser Asp Leu Asn Asp Phe Val Ser
6890 6895 6900
Asp Ala Asp Ser Thr Leu Ile Gly Asp Cys Ala Thr Val His Thr
6905 6910 6915
Ala Asn Lys Trp Asp Leu Ile Ile Ser Asp Met Tyr Asp Pro Lys
6920 6925 6930
Thr Lys Asn Val Thr Lys Glu Asn Asp Ser Lys Glu Gly Phe Phe
6935 6940 6945
Thr Tyr Ile Cys Gly Phe Ile Gln Gln Lys Leu Ala Leu Gly Gly
6950 6955 6960
Ser Val Ala Ile Lys Ile Thr Glu His Ser Trp Asn Ala Asp Leu
6965 6970 6975
Tyr Lys Leu Met Gly His Phe Ala Trp Trp Thr Ala Phe Val Thr
6980 6985 6990
Asn Val Asn Ala Ser Ser Ser Glu Ala Phe Leu Ile Gly Cys Asn
6995 7000 7005
Tyr Leu Gly Lys Pro Arg Glu Gln Ile Asp Gly Tyr Val Met His
7010 7015 7020
Ala Asn Tyr Ile Phe Trp Arg Asn Thr Asn Pro Ile Gln Leu Ser
7025 7030 7035
Ser Tyr Ser Leu Phe Asp Met Ser Lys Phe Pro Leu Lys Leu Arg
7040 7045 7050
Gly Thr Ala Val Met Ser Leu Lys Glu Gly Gln Ile Asn Asp Met
7055 7060 7065
Ile Leu Ser Leu Leu Ser Lys Gly Arg Leu Ile Ile Arg Glu Asn
7070 7075 7080
Asn Arg Val Val Ile Ser Ser Asp Val Leu Val Asn Asn
7085 7090 7095
<210> 165
<211> 1273
<212> ПРТ
<213> бетакоронавирус SARS коронавирус 2
<400> 165
Met Phe Val Phe Leu Val Leu Leu Pro Leu Val Ser Ser Gln Cys Val
1 5 10 15
Asn Leu Thr Thr Arg Thr Gln Leu Pro Pro Ala Tyr Thr Asn Ser Phe
20 25 30
Thr Arg Gly Val Tyr Tyr Pro Asp Lys Val Phe Arg Ser Ser Val Leu
35 40 45
His Ser Thr Gln Asp Leu Phe Leu Pro Phe Phe Ser Asn Val Thr Trp
50 55 60
Phe His Ala Ile His Val Ser Gly Thr Asn Gly Thr Lys Arg Phe Asp
65 70 75 80
Asn Pro Val Leu Pro Phe Asn Asp Gly Val Tyr Phe Ala Ser Thr Glu
85 90 95
Lys Ser Asn Ile Ile Arg Gly Trp Ile Phe Gly Thr Thr Leu Asp Ser
100 105 110
Lys Thr Gln Ser Leu Leu Ile Val Asn Asn Ala Thr Asn Val Val Ile
115 120 125
Lys Val Cys Glu Phe Gln Phe Cys Asn Asp Pro Phe Leu Gly Val Tyr
130 135 140
Tyr His Lys Asn Asn Lys Ser Trp Met Glu Ser Glu Phe Arg Val Tyr
145 150 155 160
Ser Ser Ala Asn Asn Cys Thr Phe Glu Tyr Val Ser Gln Pro Phe Leu
165 170 175
Met Asp Leu Glu Gly Lys Gln Gly Asn Phe Lys Asn Leu Arg Glu Phe
180 185 190
Val Phe Lys Asn Ile Asp Gly Tyr Phe Lys Ile Tyr Ser Lys His Thr
195 200 205
Pro Ile Asn Leu Val Arg Asp Leu Pro Gln Gly Phe Ser Ala Leu Glu
210 215 220
Pro Leu Val Asp Leu Pro Ile Gly Ile Asn Ile Thr Arg Phe Gln Thr
225 230 235 240
Leu Leu Ala Leu His Arg Ser Tyr Leu Thr Pro Gly Asp Ser Ser Ser
245 250 255
Gly Trp Thr Ala Gly Ala Ala Ala Tyr Tyr Val Gly Tyr Leu Gln Pro
260 265 270
Arg Thr Phe Leu Leu Lys Tyr Asn Glu Asn Gly Thr Ile Thr Asp Ala
275 280 285
Val Asp Cys Ala Leu Asp Pro Leu Ser Glu Thr Lys Cys Thr Leu Lys
290 295 300
Ser Phe Thr Val Glu Lys Gly Ile Tyr Gln Thr Ser Asn Phe Arg Val
305 310 315 320
Gln Pro Thr Glu Ser Ile Val Arg Phe Pro Asn Ile Thr Asn Leu Cys
325 330 335
Pro Phe Gly Glu Val Phe Asn Ala Thr Arg Phe Ala Ser Val Tyr Ala
340 345 350
Trp Asn Arg Lys Arg Ile Ser Asn Cys Val Ala Asp Tyr Ser Val Leu
355 360 365
Tyr Asn Ser Ala Ser Phe Ser Thr Phe Lys Cys Tyr Gly Val Ser Pro
370 375 380
Thr Lys Leu Asn Asp Leu Cys Phe Thr Asn Val Tyr Ala Asp Ser Phe
385 390 395 400
Val Ile Arg Gly Asp Glu Val Arg Gln Ile Ala Pro Gly Gln Thr Gly
405 410 415
Lys Ile Ala Asp Tyr Asn Tyr Lys Leu Pro Asp Asp Phe Thr Gly Cys
420 425 430
Val Ile Ala Trp Asn Ser Asn Asn Leu Asp Ser Lys Val Gly Gly Asn
435 440 445
Tyr Asn Tyr Leu Tyr Arg Leu Phe Arg Lys Ser Asn Leu Lys Pro Phe
450 455 460
Glu Arg Asp Ile Ser Thr Glu Ile Tyr Gln Ala Gly Ser Thr Pro Cys
465 470 475 480
Asn Gly Val Glu Gly Phe Asn Cys Tyr Phe Pro Leu Gln Ser Tyr Gly
485 490 495
Phe Gln Pro Thr Asn Gly Val Gly Tyr Gln Pro Tyr Arg Val Val Val
500 505 510
Leu Ser Phe Glu Leu Leu His Ala Pro Ala Thr Val Cys Gly Pro Lys
515 520 525
Lys Ser Thr Asn Leu Val Lys Asn Lys Cys Val Asn Phe Asn Phe Asn
530 535 540
Gly Leu Thr Gly Thr Gly Val Leu Thr Glu Ser Asn Lys Lys Phe Leu
545 550 555 560
Pro Phe Gln Gln Phe Gly Arg Asp Ile Ala Asp Thr Thr Asp Ala Val
565 570 575
Arg Asp Pro Gln Thr Leu Glu Ile Leu Asp Ile Thr Pro Cys Ser Phe
580 585 590
Gly Gly Val Ser Val Ile Thr Pro Gly Thr Asn Thr Ser Asn Gln Val
595 600 605
Ala Val Leu Tyr Gln Asp Val Asn Cys Thr Glu Val Pro Val Ala Ile
610 615 620
His Ala Asp Gln Leu Thr Pro Thr Trp Arg Val Tyr Ser Thr Gly Ser
625 630 635 640
Asn Val Phe Gln Thr Arg Ala Gly Cys Leu Ile Gly Ala Glu His Val
645 650 655
Asn Asn Ser Tyr Glu Cys Asp Ile Pro Ile Gly Ala Gly Ile Cys Ala
660 665 670
Ser Tyr Gln Thr Gln Thr Asn Ser Pro Arg Arg Ala Arg Ser Val Ala
675 680 685
Ser Gln Ser Ile Ile Ala Tyr Thr Met Ser Leu Gly Ala Glu Asn Ser
690 695 700
Val Ala Tyr Ser Asn Asn Ser Ile Ala Ile Pro Thr Asn Phe Thr Ile
705 710 715 720
Ser Val Thr Thr Glu Ile Leu Pro Val Ser Met Thr Lys Thr Ser Val
725 730 735
Asp Cys Thr Met Tyr Ile Cys Gly Asp Ser Thr Glu Cys Ser Asn Leu
740 745 750
Leu Leu Gln Tyr Gly Ser Phe Cys Thr Gln Leu Asn Arg Ala Leu Thr
755 760 765
Gly Ile Ala Val Glu Gln Asp Lys Asn Thr Gln Glu Val Phe Ala Gln
770 775 780
Val Lys Gln Ile Tyr Lys Thr Pro Pro Ile Lys Asp Phe Gly Gly Phe
785 790 795 800
Asn Phe Ser Gln Ile Leu Pro Asp Pro Ser Lys Pro Ser Lys Arg Ser
805 810 815
Phe Ile Glu Asp Leu Leu Phe Asn Lys Val Thr Leu Ala Asp Ala Gly
820 825 830
Phe Ile Lys Gln Tyr Gly Asp Cys Leu Gly Asp Ile Ala Ala Arg Asp
835 840 845
Leu Ile Cys Ala Gln Lys Phe Asn Gly Leu Thr Val Leu Pro Pro Leu
850 855 860
Leu Thr Asp Glu Met Ile Ala Gln Tyr Thr Ser Ala Leu Leu Ala Gly
865 870 875 880
Thr Ile Thr Ser Gly Trp Thr Phe Gly Ala Gly Ala Ala Leu Gln Ile
885 890 895
Pro Phe Ala Met Gln Met Ala Tyr Arg Phe Asn Gly Ile Gly Val Thr
900 905 910
Gln Asn Val Leu Tyr Glu Asn Gln Lys Leu Ile Ala Asn Gln Phe Asn
915 920 925
Ser Ala Ile Gly Lys Ile Gln Asp Ser Leu Ser Ser Thr Ala Ser Ala
930 935 940
Leu Gly Lys Leu Gln Asp Val Val Asn Gln Asn Ala Gln Ala Leu Asn
945 950 955 960
Thr Leu Val Lys Gln Leu Ser Ser Asn Phe Gly Ala Ile Ser Ser Val
965 970 975
Leu Asn Asp Ile Leu Ser Arg Leu Asp Lys Val Glu Ala Glu Val Gln
980 985 990
Ile Asp Arg Leu Ile Thr Gly Arg Leu Gln Ser Leu Gln Thr Tyr Val
995 1000 1005
Thr Gln Gln Leu Ile Arg Ala Ala Glu Ile Arg Ala Ser Ala Asn Leu
1010 1015 1020
Ala Ala Thr Lys Met Ser Glu Cys Val Leu Gly Gln Ser Lys Arg Val
1025 1030 1035 1040
Asp Phe Cys Gly Lys Gly Tyr His Leu Met Ser Phe Pro Gln Ser Ala
1045 1050 1055
Pro His Gly Val Val Phe Leu His Val Thr Tyr Val Pro Ala Gln Glu
1060 1065 1070
Lys Asn Phe Thr Thr Ala Pro Ala Ile Cys His Asp Gly Lys Ala His
1075 1080 1085
Phe Pro Arg Glu Gly Val Phe Val Ser Asn Gly Thr His Trp Phe Val
1090 1095 1100
Thr Gln Arg Asn Phe Tyr Glu Pro Gln Ile Ile Thr Thr Asp Asn Thr
1105 1110 1115 1120
Phe Val Ser Gly Asn Cys Asp Val Val Ile Gly Ile Val Asn Asn Thr
1125 1130 1135
Val Tyr Asp Pro Leu Gln Pro Glu Leu Asp Ser Phe Lys Glu Glu Leu
1140 1145 1150
Asp Lys Tyr Phe Lys Asn His Thr Ser Pro Asp Val Asp Leu Gly Asp
1155 1160 1165
Ile Ser Gly Ile Asn Ala Ser Val Val Asn Ile Gln Lys Glu Ile Asp
1170 1175 1180
Arg Leu Asn Glu Val Ala Lys Asn Leu Asn Glu Ser Leu Ile Asp Leu
1185 1190 1195 1200
Gln Glu Leu Gly Lys Tyr Glu Gln Tyr Ile Lys Trp Pro Trp Tyr Ile
1205 1210 1215
Trp Leu Gly Phe Ile Ala Gly Leu Ile Ala Ile Val Met Val Thr Ile
1220 1225 1230
Met Leu Cys Cys Met Thr Ser Cys Cys Ser Cys Leu Lys Gly Cys Cys
1235 1240 1245
Ser Cys Gly Ser Cys Cys Lys Phe Asp Glu Asp Asp Ser Glu Pro Val
1250 1255 1260
Leu Lys Gly Val Lys Leu His Tyr Thr
1265 1270
<210> 166
<211> 220
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> RBD поверхностного гликопротеина [вирус пневмонии
рынка морепродуктов в Ухане]
<400> 166
Asn Ile Thr Asn Leu Cys Pro Phe Gly Glu Val Phe Asn Ala Thr Arg
1 5 10 15
Phe Ala Ser Val Tyr Ala Trp Asn Arg Lys Arg Ile Ser Asn Cys Val
20 25 30
Ala Asp Tyr Ser Val Leu Tyr Asn Ser Ala Ser Phe Ser Thr Phe Lys
35 40 45
Cys Tyr Gly Val Ser Pro Thr Lys Leu Asn Asp Leu Cys Phe Thr Asn
50 55 60
Val Tyr Ala Asp Ser Phe Val Ile Arg Gly Asp Glu Val Arg Gln Ile
65 70 75 80
Ala Pro Gly Gln Thr Gly Lys Ile Ala Asp Tyr Asn Tyr Lys Leu Pro
85 90 95
Asp Asp Phe Thr Gly Cys Val Ile Ala Trp Asn Ser Asn Asn Leu Asp
100 105 110
Ser Lys Val Gly Gly Asn Tyr Asn Tyr Leu Tyr Arg Leu Phe Arg Lys
115 120 125
Ser Asn Leu Lys Pro Phe Glu Arg Asp Ile Ser Thr Glu Ile Tyr Gln
130 135 140
Ala Gly Ser Thr Pro Cys Asn Gly Val Glu Gly Phe Asn Cys Tyr Phe
145 150 155 160
Pro Leu Gln Ser Tyr Gly Phe Gln Pro Thr Asn Gly Val Gly Tyr Gln
165 170 175
Pro Tyr Arg Val Val Val Leu Ser Phe Glu Leu Leu His Ala Pro Ala
180 185 190
Thr Val Cys Gly Pro Lys Lys Ser Thr Asn Leu Val Lys Asn Lys Cys
195 200 205
Val Asn Phe Asn Phe Asn Gly Leu Thr Gly Thr Gly
210 215 220
<210> 167
<211> 72
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Рецептор-связывающий мотив (RBM) в поверхностном
гликопротеине RBD [вирус пневмонии рынка морепродуктов в
Ухане]
<400> 167
Asn Ser Asn Asn Leu Asp Ser Lys Val Gly Gly Asn Tyr Asn Tyr Leu
1 5 10 15
Tyr Arg Leu Phe Arg Lys Ser Asn Leu Lys Pro Phe Glu Arg Asp Ile
20 25 30
Ser Thr Glu Ile Tyr Gln Ala Gly Ser Thr Pro Cys Asn Gly Val Glu
35 40 45
Gly Phe Asn Cys Tyr Phe Pro Leu Gln Ser Tyr Gly Phe Gln Pro Thr
50 55 60
Asn Gly Val Gly Tyr Gln Pro Tyr
65 70
<210> 168
<211> 107
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> VL (VK) S309-v13 mAb SARS-CoV-2
<400> 168
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Thr Val Ser Ser Thr
20 25 30
Ser Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln His Asp Thr Ser Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 169
<211> 7
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> CDRL1 S309-v13 mAb SARS-CoV-2
<400> 169
Gln Thr Val Ser Ser Thr Ser
1 5
<210> 170
<211> 3
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> CDRL2 S309-v13 mAb SARS-CoV-2
<400> 170
Gly Ala Ser
1
<210> 171
<211> 8
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> CDRL3 S309-v13 mAb SARS-CoV-2
<400> 171
Gln Gln His Asp Thr Ser Leu Thr
1 5
<210> 172
<211> 127
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> VH S309-v2.9 mAb SARS-CoV-2
<400> 172
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Phe Ile Ser Thr Tyr Asn Ala Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr
65 70 75 80
Met Glu Leu Arg Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Thr Arg Gly Ala Phe Phe Gly Glu Ser Leu Ile Gly
100 105 110
Gly Phe Asp Asn Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 173
<211> 330
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> CH1-CH3 G1m17 SARS-CoV-2; IgG1*01 LS
<400> 173
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys
1 5 10 15
Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr
65 70 75 80
Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys
100 105 110
Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro
115 120 125
Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys
130 135 140
Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp
145 150 155 160
Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu
165 170 175
Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu
180 185 190
His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn
195 200 205
Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly
210 215 220
Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu
225 230 235 240
Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr
245 250 255
Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn
260 265 270
Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe
275 280 285
Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn
290 295 300
Val Phe Ser Cys Ser Val Leu His Glu Ala Leu His Ser His Tyr Thr
305 310 315 320
Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
325 330
<210> 174
<211> 107
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> CL (Ck) mAb SARS-CoV-2 IgKC*01 k1m3
<400> 174
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
1 5 10 15
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
20 25 30
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
35 40 45
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
50 55 60
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
65 70 75 80
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
85 90 95
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
100 105
<210> 175
<211> 330
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> CH1-CH3 G1m17 SARS-CoV-2; IgG1*01 LS GAALIE
<400> 175
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys
1 5 10 15
Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr
65 70 75 80
Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys
100 105 110
Pro Ala Pro Glu Leu Leu Ala Gly Pro Ser Val Phe Leu Phe Pro Pro
115 120 125
Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys
130 135 140
Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp
145 150 155 160
Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu
165 170 175
Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu
180 185 190
His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn
195 200 205
Lys Ala Leu Pro Leu Pro Glu Glu Lys Thr Ile Ser Lys Ala Lys Gly
210 215 220
Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu
225 230 235 240
Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr
245 250 255
Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn
260 265 270
Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe
275 280 285
Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn
290 295 300
Val Phe Ser Cys Ser Val Leu His Glu Ala Leu His Ser His Tyr Thr
305 310 315 320
Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
325 330
<210> 176
<211> 123
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> VH S300-v2.10 mAb SARS-CoV-2
<400> 176
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Leu
35 40 45
Gly Phe Val Asn Ala Tyr Ser Gly Ala Thr Arg Tyr Ala Gln Lys Tyr
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Arg Leu Arg Pro Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Arg Pro Ser His Glu Phe Ala Met Tyr Phe Phe Asp Asn
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 177
<211> 123
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> VH S300-v2.11 mAb SARS-CoV-2
<400> 177
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Pro Glu Trp Leu
35 40 45
Gly Phe Val Gln Gly Tyr Ser Gly Ala Thr Arg Tyr Ala Gln Lys Tyr
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Arg Leu Arg Pro Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Arg Pro Ser His Glu Phe Ala Met Tyr Phe Phe Asp Asn
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 178
<211> 124
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> VH S315-v1 mAb SARS-CoV-2
<400> 178
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asn Tyr
20 25 30
Trp Met Thr Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Asn Ile Lys Gln Asp Gly Ser Glu Lys Tyr Tyr Val Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Leu Trp Trp Asn Asp Gln Ala His Tyr Tyr Gly Met Asp
100 105 110
Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 179
<211> 8
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> CDRH1 S315-v1 mAb SARS-CoV-2
<400> 179
Gly Phe Thr Phe Ser Asn Tyr Trp
1 5
<210> 180
<211> 8
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> CDRH2 S315-v1 mAb SARS-CoV-2
<400> 180
Ile Lys Gln Asp Gly Ser Glu Lys
1 5
<210> 181
<211> 17
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> CDRH3 S315-v1 mAb SARS-CoV-2
<400> 181
Ala Arg Asp Leu Trp Trp Asn Asp Gln Ala His Tyr Tyr Gly Met Asp
1 5 10 15
Val
<210> 182
<211> 106
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> VL S315-v1 mAb SARS-CoV-2
<400> 182
Ser Tyr Glu Leu Thr Gln Pro Pro Ser Val Ser Val Ser Pro Gly Gln
1 5 10 15
Thr Ala Arg Ile Thr Cys Ser Gly Asp Ala Phe Pro Asn Gln Tyr Ala
20 25 30
Tyr Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Met Leu Ile Tyr
35 40 45
Lys Asp Ser Glu Arg Pro Ser Gly Ile Pro Glu Arg Phe Phe Gly Ser
50 55 60
Ser Ser Gly Thr Thr Val Thr Leu Thr Ile Arg Gly Val Gln Ala Glu
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Ala Asp Ser Ser Gly Thr Val
85 90 95
Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105
<210> 183
<211> 6
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> CDRL1 S315-v1 mAb SARS-CoV-2
<400> 183
Ala Phe Pro Asn Gln Tyr
1 5
<210> 184
<211> 3
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> CDRL2 S315-v1 mAb SARS-CoV-2
<400> 184
Lys Asp Ser
1
<210> 185
<211> 9
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> CDRL3 S315-v1 mAb SARS-CoV-2
<400> 185
Gln Ser Ala Asp Ser Ser Gly Thr Val
1 5
<210> 186
<211> 373
<212> ДНК
<213> Искусственная последовательность
<220>
<223> VH S315-v1 mAb SARS-CoV-2
<400> 186
gaggtgcagc tggtggagtc tgggggaggc ttggtccagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt cacctttagt aattattgga tgacctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtggccaac ataaagcaag atggaagtga gaaatactat 180
gtggactctg tgaagggccg attcaccatc tccagagaca acgccaagaa ctcactgtat 240
ctgcaaatga acagcctgag agccgaggac acggctgtgt attactgtgc gagagatctt 300
tggtggaacg accaggctca ctactacggt atggacgtct ggggccaagg gaccacggtc 360
accgtctcct cag 373
<210> 187
<211> 319
<212> ДНК
<213> Искусственная последовательность
<220>
<223> VL S315-v1 mAb SARS-CoV-2
<400> 187
tcctatgagc tgacacagcc accctcggtg tcagtgtccc caggacagac ggccaggatc 60
acctgctctg gagatgcatt cccaaaccaa tatgcttatt ggtaccagca gaagccaggc 120
caggcccctg tgatgctgat ctataaagac agtgagaggc cctcagggat ccctgagcga 180
ttctttggct ccagctcagg gacaacagtc acgttgacca tcagaggagt ccaggcagaa 240
gacgaggctg actattactg tcaatcagca gacagcagtg gtaccgtgtt cggcggaggg 300
accaagctga ccgtcctag 319
<210> 188
<211> 372
<212> ДНК
<213> Искусственная последовательность
<220>
<223> VH S315-v1 mAb SARS-CoV-2 (nt - кодон-оптимизированная)
<400> 188
gaagtgcagc ttgtcgagag cggcggaggc ctcgttcagc caggtgggag tctccgtctt 60
tcatgcgccg cttcaggatt tacgttctcc aactactgga tgacatgggt gaggcaggca 120
cctgggaagg ggctggagtg ggtggctaac atcaagcagg acggatctga aaaatattat 180
gtagattctg tgaaggggcg gtttaccatc tcaagggata atgccaaaaa ctctttgtat 240
ttacagatga actctcttcg agccgaggac accgccgttt actactgtgc ccgagatcta 300
tggtggaatg accaggctca ctattatgga atggacgtgt ggggccaggg tactaccgtt 360
accgtctcct ca 372
<210> 189
<211> 318
<212> ДНК
<213> Искусственная последовательность
<220>
<223> VL S315-v1 mAb SARS-CoV-2 (nt- кодон-оптимизированная)
<400> 189
tcttacgagc tcacccagcc accctcagtg tcagtgagcc ctggccaaac agctcgcatc 60
acctgttcag gtgacgcctt tccaaatcag tacgcctact ggtatcagca gaaacccggc 120
caggcacccg ttatgctcat ctacaaagat tctgagcggc catccggtat ccccgaacgc 180
tttttcggaa gctccagtgg gactacagtt acacttacta tccggggagt gcaagctgaa 240
gatgaggccg actattattg ccagagcgca gactcctcag gcacagtgtt tgggggcggg 300
actaaactaa ctgtgctg 318
<210> 190
<211> 106
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> VL S315-v2 mAb SARS-CoV-2
<400> 190
Ser Tyr Glu Leu Thr Gln Pro Pro Ser Val Ser Val Ser Pro Gly Gln
1 5 10 15
Thr Ala Arg Ile Thr Cys Ser Gly Asp Ala Phe Pro Asn Gln Tyr Ala
20 25 30
Tyr Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Met Leu Ile Tyr
35 40 45
Lys Asp Ser Glu Arg Pro Ser Gly Ile Pro Glu Arg Phe Phe Gly Ser
50 55 60
Ser Ser Gly Thr Thr Val Thr Leu Thr Ile Ser Gly Val Gln Ala Glu
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Ala Asp Ser Ser Gly Thr Val
85 90 95
Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105
<210> 191
<211> 319
<212> ДНК
<213> Искусственная последовательность
<220>
<223> VL S315-v2 mAb SARS-CoV-2
<400> 191
tcctatgagc tgacacagcc accctcggtg tcagtgtccc caggacagac ggccaggatc 60
acctgctctg gagatgcatt cccaaaccaa tatgcttatt ggtaccagca gaagccaggc 120
caggcccctg tgatgctgat ctataaagac agtgagaggc cctcagggat ccctgagcga 180
ttctttggct ccagctcagg gacaacagtc acgttgacca tcagtggagt ccaggcagaa 240
gacgaggctg actattactg tcaatcagca gacagcagtg gtaccgtgtt cggcggaggg 300
accaagctga ccgtcctag 319
<210> 192
<211> 318
<212> ДНК
<213> Искусственная последовательность
<220>
<223> VL S315-v2 mAb SARS-CoV-2 (nt - кодон-оптимизированная)
<400> 192
tcctacgagc tcacccagcc cccctcagtc tctgtgtctc ctggacagac agccagaatc 60
acctgctcgg gagatgcttt tcccaaccaa tacgcctact ggtaccaaca gaaaccaggt 120
caggcgcctg tcatgctgat ttataaagac tcagagcggc cttcaggaat tcccgaaaga 180
ttcttcggga gttcaagcgg aactaccgtg accttaacca taagcggggt gcaggccgaa 240
gatgaagcag actattattg ccagagtgcc gatagtagtg gcacagtctt tggggggggg 300
acaaagctga cagtactc 318
<210> 193
<211> 106
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> CL mAb IgLC*01 SARS-CoV-2
<400> 193
Gly Gln Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser
1 5 10 15
Glu Glu Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp
20 25 30
Phe Tyr Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro
35 40 45
Val Lys Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn
50 55 60
Lys Tyr Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys
65 70 75 80
Ser His Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val
85 90 95
Glu Lys Thr Val Ala Pro Thr Glu Cys Ser
100 105
<210> 194
<211> 124
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> VH S315-v3 mAb SARS-CoV-2
<400> 194
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asn Tyr
20 25 30
Phe Met Thr Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Asn Ile Lys Gln Asp Gly Ser Glu Lys Tyr Tyr Val Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Leu Trp Trp Asn Asp Gln Ala His Tyr Tyr Gly Met Asp
100 105 110
Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 195
<211> 8
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> CDRH1 S315-v3 mAb SARS-CoV-2
<400> 195
Gly Phe Thr Phe Ser Asn Tyr Phe
1 5
<210> 196
<211> 124
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> VH S315-v4 mAb SARS-CoV-2
<400> 196
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asn Tyr
20 25 30
Trp Met Thr Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Asn Ile Lys Gln Asp Ala Ser Glu Lys Tyr Tyr Val Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Leu Trp Trp Asn Asp Gln Ala His Tyr Tyr Gly Met Asp
100 105 110
Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 197
<211> 8
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> CDRH2 S315-v4 mAb SARS-CoV-2
<400> 197
Ile Lys Gln Asp Ala Ser Glu Lys
1 5
<210> 198
<211> 124
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> VH S315-v5 mAb SARS-CoV-2
<400> 198
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asn Tyr
20 25 30
Trp Met Thr Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Asn Ile Lys Gln Glu Gly Ser Glu Lys Tyr Tyr Val Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Leu Trp Trp Asn Asp Gln Ala His Tyr Tyr Gly Met Asp
100 105 110
Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 199
<211> 8
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> CDRH2 S315-v5 mAb SARS-CoV-2
<400> 199
Ile Lys Gln Glu Gly Ser Glu Lys
1 5
<210> 200
<211> 124
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> VH S315-v6 mAb SARS-CoV-2
<400> 200
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asn Tyr
20 25 30
Trp Met Thr Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Asn Ile Lys Gln Asp Gly Ser Glu Lys Tyr Tyr Val Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Leu Phe Trp Asn Asp Gln Ala His Tyr Tyr Gly Met Asp
100 105 110
Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 201
<211> 17
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> CDRH3 S315-v6 mAb SARS-CoV-2
<400> 201
Ala Arg Asp Leu Phe Trp Asn Asp Gln Ala His Tyr Tyr Gly Met Asp
1 5 10 15
Val
<210> 202
<211> 124
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> VH S315-v7 mAb SARS-CoV-2
<400> 202
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asn Tyr
20 25 30
Trp Met Thr Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Asn Ile Lys Gln Asp Gly Ser Glu Lys Tyr Tyr Val Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Leu Trp Phe Asn Asp Gln Ala His Tyr Tyr Gly Met Asp
100 105 110
Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 203
<211> 17
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> CDRH3 S315-v7 mAb SARS-CoV-2
<400> 203
Ala Arg Asp Leu Trp Phe Asn Asp Gln Ala His Tyr Tyr Gly Met Asp
1 5 10 15
Val
<210> 204
<211> 230
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Тяжелая цепь IgHG1*01 Fd SARS-CoV-2
<400> 204
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Thr Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr
65 70 75 80
Met Glu Leu Arg Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Thr Arg Gly Ala Trp Phe Gly Glu Ser Leu Ile Gly
100 105 110
Gly Phe Asp Asn Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala
115 120 125
Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser
130 135 140
Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe
145 150 155 160
Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly
165 170 175
Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu
180 185 190
Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr
195 200 205
Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg
210 215 220
Val Glu Pro Lys Ser Cys
225 230
<210> 205
<211> 214
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Легкая цепь IgKC*01 SARS-CoV-2
<400> 205
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Thr Val Ser Ser Thr
20 25 30
Ser Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln His Asp Thr Ser Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 206
<211> 18
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Линкер
<400> 206
Gly Ser Thr Ser Gly Ser Gly Lys Pro Gly Ser Gly Glu Gly Ser Thr
1 5 10 15
Lys Gly
<210> 207
<211> 10
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Линкер
<400> 207
Gly Ser Gly Lys Pro Gly Ser Gly Glu Gly
1 5 10
<210> 208
<211> 8
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Линкер
<400> 208
Gly Lys Pro Gly Ser Gly Glu Gly
1 5
<210> 209
<211> 8
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Линкер
<400> 209
Ser Gly Lys Pro Gly Ser Gly Glu
1 5
<210> 210
<211> 6
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Линкер
<220>
<221> ВАРИАНТ
<222> 1
<223> Xaa представляет собой положительно заряженную аминокислоту
и Z представляет собой глицин или отрицательно
заряженную аминокислоту
<220>
<221> ВАРИАНТ
<222> (3)...(5)
<223> Xaa представляет собой Gly или Ser
<400> 210
Xaa Pro Xaa Xaa Xaa Glx
1 5
<210> 211
<211> 121
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Линкер
<220>
<221> ВАРИАНТ
<222> (2)...(10)
<223> любая одна или все аминокислоты 2-10 могут либо присутствовать,
либо отсутствовать
<220>
<221> ВАРИАНТ
<222> (12)...(121)
<223> любая одна или все аминокислоты 12-121 могут либо
присутствовать, либо отсутствовать
<400> 211
Gly Gly Gly Gly Gly Gly Gly Gly Gly Gly Ser Gly Gly Gly Gly Gly
1 5 10 15
Gly Gly Gly Gly Gly Ser Gly Gly Gly Gly Gly Gly Gly Gly Gly Gly
20 25 30
Ser Gly Gly Gly Gly Gly Gly Gly Gly Gly Gly Ser Gly Gly Gly Gly
35 40 45
Gly Gly Gly Gly Gly Gly Ser Gly Gly Gly Gly Gly Gly Gly Gly Gly
50 55 60
Gly Ser Gly Gly Gly Gly Gly Gly Gly Gly Gly Gly Ser Gly Gly Gly
65 70 75 80
Gly Gly Gly Gly Gly Gly Gly Ser Gly Gly Gly Gly Gly Gly Gly Gly
85 90 95
Gly Gly Ser Gly Gly Gly Gly Gly Gly Gly Gly Gly Gly Ser Gly Gly
100 105 110
Gly Gly Gly Gly Gly Gly Gly Gly Ser
115 120
<210> 212
<211> 15
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Линкер
<400> 212
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
1 5 10 15
<210> 213
<211> 50
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Линкер
<400> 213
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
1 5 10 15
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
20 25 30
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
35 40 45
Gly Ser
50
<210> 214
<211> 18
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Линкер
<400> 214
Gly Ser Thr Ser Gly Gly Gly Ser Gly Gly Gly Ser Gly Gly Gly Gly
1 5 10 15
Ser Ser
<210> 215
<211> 14
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Линкер
<400> 215
Glu Gly Lys Ser Ser Gly Ser Gly Ser Glu Ser Lys Val Asp
1 5 10
<210> 216
<211> 18
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Линкер
<400> 216
Lys Glu Ser Gly Ser Val Ser Ser Glu Gln Leu Ala Gln Phe Arg Ser
1 5 10 15
Leu Asp
<210> 217
<211> 5
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Линкер
<400> 217
Gly Gly Gly Gly Ser
1 5
<210> 218
<211> 494
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
S309-scFab (H-L) SARS-CoV-2
<400> 218
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Thr Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr
65 70 75 80
Met Glu Leu Arg Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Thr Arg Gly Ala Trp Phe Gly Glu Ser Leu Ile Gly
100 105 110
Gly Phe Asp Asn Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala
115 120 125
Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser
130 135 140
Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe
145 150 155 160
Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly
165 170 175
Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu
180 185 190
Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr
195 200 205
Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg
210 215 220
Val Glu Pro Lys Ser Cys Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
225 230 235 240
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
245 250 255
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
260 265 270
Gly Gly Ser Gly Gly Gly Gly Ser Glu Ile Val Leu Thr Gln Ser Pro
275 280 285
Gly Thr Leu Ser Leu Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg
290 295 300
Ala Ser Gln Thr Val Ser Ser Thr Ser Leu Ala Trp Tyr Gln Gln Lys
305 310 315 320
Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr Gly Ala Ser Ser Arg Ala
325 330 335
Thr Gly Ile Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe
340 345 350
Thr Leu Thr Ile Ser Arg Leu Glu Pro Glu Asp Phe Ala Val Tyr Tyr
355 360 365
Cys Gln Gln His Asp Thr Ser Leu Thr Phe Gly Gly Gly Thr Lys Val
370 375 380
Glu Ile Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro
385 390 395 400
Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu
405 410 415
Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn
420 425 430
Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser
435 440 445
Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala
450 455 460
Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly
465 470 475 480
Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
485 490
<210> 219
<211> 494
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> S309-scFab (L-H) SARS-CoV-2
<400> 219
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Thr Val Ser Ser Thr
20 25 30
Ser Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln His Asp Thr Ser Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
210 215 220
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
225 230 235 240
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
245 250 255
Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln Leu Val Gln Ser Gly
260 265 270
Ala Glu Val Lys Lys Pro Gly Ala Ser Val Lys Val Ser Cys Lys Ala
275 280 285
Ser Gly Tyr Pro Phe Thr Ser Tyr Gly Ile Ser Trp Val Arg Gln Ala
290 295 300
Pro Gly Gln Gly Leu Glu Trp Met Gly Trp Ile Ser Thr Tyr Asn Gly
305 310 315 320
Asn Thr Asn Tyr Ala Gln Lys Phe Gln Gly Arg Val Thr Met Thr Thr
325 330 335
Asp Thr Ser Thr Thr Thr Gly Tyr Met Glu Leu Arg Arg Leu Arg Ser
340 345 350
Asp Asp Thr Ala Val Tyr Tyr Cys Ala Arg Asp Tyr Thr Arg Gly Ala
355 360 365
Trp Phe Gly Glu Ser Leu Ile Gly Gly Phe Asp Asn Trp Gly Gln Gly
370 375 380
Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe
385 390 395 400
Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu
405 410 415
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
420 425 430
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
435 440 445
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
450 455 460
Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro
465 470 475 480
Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys
485 490
<210> 220
<211> 249
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> S309-scFv (VH-VL) SARS-CoV-2
<400> 220
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Thr Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr
65 70 75 80
Met Glu Leu Arg Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Thr Arg Gly Ala Trp Phe Gly Glu Ser Leu Ile Gly
100 105 110
Gly Phe Asp Asn Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Ile
130 135 140
Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly Glu Arg
145 150 155 160
Ala Thr Leu Ser Cys Arg Ala Ser Gln Thr Val Ser Ser Thr Ser Leu
165 170 175
Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr
180 185 190
Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser Gly Ser
195 200 205
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu Pro Glu
210 215 220
Asp Phe Ala Val Tyr Tyr Cys Gln Gln His Asp Thr Ser Leu Thr Phe
225 230 235 240
Gly Gly Gly Thr Lys Val Glu Ile Lys
245
<210> 221
<211> 249
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> S309-scFv (VL-VH) SARS-CoV-2
<400> 221
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Thr Val Ser Ser Thr
20 25 30
Ser Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln His Asp Thr Ser Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Gly Gly Gly Gly Ser
100 105 110
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln Leu Val Gln
115 120 125
Ser Gly Ala Glu Val Lys Lys Pro Gly Ala Ser Val Lys Val Ser Cys
130 135 140
Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr Gly Ile Ser Trp Val Arg
145 150 155 160
Gln Ala Pro Gly Gln Gly Leu Glu Trp Met Gly Trp Ile Ser Thr Tyr
165 170 175
Asn Gly Asn Thr Asn Tyr Ala Gln Lys Phe Gln Gly Arg Val Thr Met
180 185 190
Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr Met Glu Leu Arg Arg Leu
195 200 205
Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys Ala Arg Asp Tyr Thr Arg
210 215 220
Gly Ala Trp Phe Gly Glu Ser Leu Ile Gly Gly Phe Asp Asn Trp Gly
225 230 235 240
Gln Gly Thr Leu Val Thr Val Ser Ser
245
<210> 222
<211> 518
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> S309-scFv (VH-VL)-(VH-VL) SARS-CoV-2
<400> 222
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Thr Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr
65 70 75 80
Met Glu Leu Arg Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Thr Arg Gly Ala Trp Phe Gly Glu Ser Leu Ile Gly
100 105 110
Gly Phe Asp Asn Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Ile
130 135 140
Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly Glu Arg
145 150 155 160
Ala Thr Leu Ser Cys Arg Ala Ser Gln Thr Val Ser Ser Thr Ser Leu
165 170 175
Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr
180 185 190
Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser Gly Ser
195 200 205
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu Pro Glu
210 215 220
Asp Phe Ala Val Tyr Tyr Cys Gln Gln His Asp Thr Ser Leu Thr Phe
225 230 235 240
Gly Gly Gly Thr Lys Val Glu Ile Lys Gly Gly Gly Gly Ser Gly Gly
245 250 255
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln
260 265 270
Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala Ser Val Lys
275 280 285
Val Ser Cys Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr Gly Ile Ser
290 295 300
Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met Gly Trp Ile
305 310 315 320
Ser Thr Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Phe Gln Gly Arg
325 330 335
Val Thr Met Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr Met Glu Leu
340 345 350
Arg Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys Ala Arg Asp
355 360 365
Tyr Thr Arg Gly Ala Trp Phe Gly Glu Ser Leu Ile Gly Gly Phe Asp
370 375 380
Asn Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
385 390 395 400
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Ile Val Leu Thr
405 410 415
Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly Glu Arg Ala Thr Leu
420 425 430
Ser Cys Arg Ala Ser Gln Thr Val Ser Ser Thr Ser Leu Ala Trp Tyr
435 440 445
Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr Gly Ala Ser
450 455 460
Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly
465 470 475 480
Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu Pro Glu Asp Phe Ala
485 490 495
Val Tyr Tyr Cys Gln Gln His Asp Thr Ser Leu Thr Phe Gly Gly Gly
500 505 510
Thr Lys Val Glu Ile Lys
515
<210> 223
<211> 518
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> S309-scFv-(VH-VL)-(VL-VH) SARS-CoV-2
<400> 223
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Thr Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr
65 70 75 80
Met Glu Leu Arg Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Thr Arg Gly Ala Trp Phe Gly Glu Ser Leu Ile Gly
100 105 110
Gly Phe Asp Asn Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Ile
130 135 140
Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly Glu Arg
145 150 155 160
Ala Thr Leu Ser Cys Arg Ala Ser Gln Thr Val Ser Ser Thr Ser Leu
165 170 175
Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr
180 185 190
Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser Gly Ser
195 200 205
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu Pro Glu
210 215 220
Asp Phe Ala Val Tyr Tyr Cys Gln Gln His Asp Thr Ser Leu Thr Phe
225 230 235 240
Gly Gly Gly Thr Lys Val Glu Ile Lys Gly Gly Gly Gly Ser Gly Gly
245 250 255
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Ile Val
260 265 270
Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly Glu Arg Ala
275 280 285
Thr Leu Ser Cys Arg Ala Ser Gln Thr Val Ser Ser Thr Ser Leu Ala
290 295 300
Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr Gly
305 310 315 320
Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser Gly Ser Gly
325 330 335
Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu Pro Glu Asp
340 345 350
Phe Ala Val Tyr Tyr Cys Gln Gln His Asp Thr Ser Leu Thr Phe Gly
355 360 365
Gly Gly Thr Lys Val Glu Ile Lys Gly Gly Gly Gly Ser Gly Gly Gly
370 375 380
Gly Ser Gly Gly Gly Gly Ser Gln Val Gln Leu Val Gln Ser Gly Ala
385 390 395 400
Glu Val Lys Lys Pro Gly Ala Ser Val Lys Val Ser Cys Lys Ala Ser
405 410 415
Gly Tyr Pro Phe Thr Ser Tyr Gly Ile Ser Trp Val Arg Gln Ala Pro
420 425 430
Gly Gln Gly Leu Glu Trp Met Gly Trp Ile Ser Thr Tyr Asn Gly Asn
435 440 445
Thr Asn Tyr Ala Gln Lys Phe Gln Gly Arg Val Thr Met Thr Thr Asp
450 455 460
Thr Ser Thr Thr Thr Gly Tyr Met Glu Leu Arg Arg Leu Arg Ser Asp
465 470 475 480
Asp Thr Ala Val Tyr Tyr Cys Ala Arg Asp Tyr Thr Arg Gly Ala Trp
485 490 495
Phe Gly Glu Ser Leu Ile Gly Gly Phe Asp Asn Trp Gly Gln Gly Thr
500 505 510
Leu Val Thr Val Ser Ser
515
<210> 224
<211> 518
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> S309-scFv-(VL-VH)-(VH-VL) SARS-CoV-2
<400> 224
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Thr Val Ser Ser Thr
20 25 30
Ser Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln His Asp Thr Ser Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Gly Gly Gly Gly Ser
100 105 110
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln Leu Val Gln
115 120 125
Ser Gly Ala Glu Val Lys Lys Pro Gly Ala Ser Val Lys Val Ser Cys
130 135 140
Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr Gly Ile Ser Trp Val Arg
145 150 155 160
Gln Ala Pro Gly Gln Gly Leu Glu Trp Met Gly Trp Ile Ser Thr Tyr
165 170 175
Asn Gly Asn Thr Asn Tyr Ala Gln Lys Phe Gln Gly Arg Val Thr Met
180 185 190
Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr Met Glu Leu Arg Arg Leu
195 200 205
Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys Ala Arg Asp Tyr Thr Arg
210 215 220
Gly Ala Trp Phe Gly Glu Ser Leu Ile Gly Gly Phe Asp Asn Trp Gly
225 230 235 240
Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly
245 250 255
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln
260 265 270
Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala Ser Val Lys
275 280 285
Val Ser Cys Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr Gly Ile Ser
290 295 300
Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met Gly Trp Ile
305 310 315 320
Ser Thr Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Phe Gln Gly Arg
325 330 335
Val Thr Met Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr Met Glu Leu
340 345 350
Arg Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys Ala Arg Asp
355 360 365
Tyr Thr Arg Gly Ala Trp Phe Gly Glu Ser Leu Ile Gly Gly Phe Asp
370 375 380
Asn Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
385 390 395 400
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Ile Val Leu Thr
405 410 415
Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly Glu Arg Ala Thr Leu
420 425 430
Ser Cys Arg Ala Ser Gln Thr Val Ser Ser Thr Ser Leu Ala Trp Tyr
435 440 445
Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr Gly Ala Ser
450 455 460
Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly
465 470 475 480
Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu Pro Glu Asp Phe Ala
485 490 495
Val Tyr Tyr Cys Gln Gln His Asp Thr Ser Leu Thr Phe Gly Gly Gly
500 505 510
Thr Lys Val Glu Ile Lys
515
<210> 225
<211> 518
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
S309-scFv-(VL-VH)-(VL-VH) SARS-CoV-2
<400> 225
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Thr Val Ser Ser Thr
20 25 30
Ser Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln His Asp Thr Ser Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Gly Gly Gly Gly Ser
100 105 110
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln Leu Val Gln
115 120 125
Ser Gly Ala Glu Val Lys Lys Pro Gly Ala Ser Val Lys Val Ser Cys
130 135 140
Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr Gly Ile Ser Trp Val Arg
145 150 155 160
Gln Ala Pro Gly Gln Gly Leu Glu Trp Met Gly Trp Ile Ser Thr Tyr
165 170 175
Asn Gly Asn Thr Asn Tyr Ala Gln Lys Phe Gln Gly Arg Val Thr Met
180 185 190
Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr Met Glu Leu Arg Arg Leu
195 200 205
Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys Ala Arg Asp Tyr Thr Arg
210 215 220
Gly Ala Trp Phe Gly Glu Ser Leu Ile Gly Gly Phe Asp Asn Trp Gly
225 230 235 240
Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly
245 250 255
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Ile Val
260 265 270
Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly Glu Arg Ala
275 280 285
Thr Leu Ser Cys Arg Ala Ser Gln Thr Val Ser Ser Thr Ser Leu Ala
290 295 300
Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr Gly
305 310 315 320
Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser Gly Ser Gly
325 330 335
Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu Pro Glu Asp
340 345 350
Phe Ala Val Tyr Tyr Cys Gln Gln His Asp Thr Ser Leu Thr Phe Gly
355 360 365
Gly Gly Thr Lys Val Glu Ile Lys Gly Gly Gly Gly Ser Gly Gly Gly
370 375 380
Gly Ser Gly Gly Gly Gly Ser Gln Val Gln Leu Val Gln Ser Gly Ala
385 390 395 400
Glu Val Lys Lys Pro Gly Ala Ser Val Lys Val Ser Cys Lys Ala Ser
405 410 415
Gly Tyr Pro Phe Thr Ser Tyr Gly Ile Ser Trp Val Arg Gln Ala Pro
420 425 430
Gly Gln Gly Leu Glu Trp Met Gly Trp Ile Ser Thr Tyr Asn Gly Asn
435 440 445
Thr Asn Tyr Ala Gln Lys Phe Gln Gly Arg Val Thr Met Thr Thr Asp
450 455 460
Thr Ser Thr Thr Thr Gly Tyr Met Glu Leu Arg Arg Leu Arg Ser Asp
465 470 475 480
Asp Thr Ala Val Tyr Tyr Cys Ala Arg Asp Tyr Thr Arg Gly Ala Trp
485 490 495
Phe Gly Glu Ser Leu Ile Gly Gly Phe Asp Asn Trp Gly Gln Gly Thr
500 505 510
Leu Val Thr Val Ser Ser
515
<210> 226
<211> 494
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность S309-scFab-(H-L)
v1.1 SARS-CoV-2
<400> 226
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Thr Tyr Gln Gly Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr
65 70 75 80
Met Glu Leu Arg Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Thr Arg Gly Ala Trp Phe Gly Glu Ser Leu Ile Gly
100 105 110
Gly Phe Asp Asn Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala
115 120 125
Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser
130 135 140
Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe
145 150 155 160
Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly
165 170 175
Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu
180 185 190
Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr
195 200 205
Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg
210 215 220
Val Glu Pro Lys Ser Cys Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
225 230 235 240
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
245 250 255
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
260 265 270
Gly Gly Ser Gly Gly Gly Gly Ser Glu Ile Val Leu Thr Gln Ser Pro
275 280 285
Gly Thr Leu Ser Leu Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg
290 295 300
Ala Ser Gln Thr Val Ser Ser Thr Ser Leu Ala Trp Tyr Gln Gln Lys
305 310 315 320
Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr Gly Ala Ser Ser Arg Ala
325 330 335
Thr Gly Ile Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe
340 345 350
Thr Leu Thr Ile Ser Arg Leu Glu Pro Glu Asp Phe Ala Val Tyr Tyr
355 360 365
Cys Gln Gln His Asp Thr Ser Leu Thr Phe Gly Gly Gly Thr Lys Val
370 375 380
Glu Ile Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro
385 390 395 400
Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu
405 410 415
Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn
420 425 430
Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser
435 440 445
Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala
450 455 460
Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly
465 470 475 480
Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
485 490
<210> 227
<211> 494
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность S309-scFab-(L-H)
v1.1 SARS-CoV-2
<400> 227
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Thr Val Ser Ser Thr
20 25 30
Ser Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln His Asp Thr Ser Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
210 215 220
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
225 230 235 240
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
245 250 255
Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln Leu Val Gln Ser Gly
260 265 270
Ala Glu Val Lys Lys Pro Gly Ala Ser Val Lys Val Ser Cys Lys Ala
275 280 285
Ser Gly Tyr Pro Phe Thr Ser Tyr Gly Ile Ser Trp Val Arg Gln Ala
290 295 300
Pro Gly Gln Gly Leu Glu Trp Met Gly Trp Ile Ser Thr Tyr Gln Gly
305 310 315 320
Asn Thr Asn Tyr Ala Gln Lys Phe Gln Gly Arg Val Thr Met Thr Thr
325 330 335
Asp Thr Ser Thr Thr Thr Gly Tyr Met Glu Leu Arg Arg Leu Arg Ser
340 345 350
Asp Asp Thr Ala Val Tyr Tyr Cys Ala Arg Asp Tyr Thr Arg Gly Ala
355 360 365
Trp Phe Gly Glu Ser Leu Ile Gly Gly Phe Asp Asn Trp Gly Gln Gly
370 375 380
Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe
385 390 395 400
Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu
405 410 415
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
420 425 430
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
435 440 445
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
450 455 460
Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro
465 470 475 480
Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys
485 490
<210> 228
<211> 249
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность S309-scFv-(VH-VL)
v1.1 SARS-CoV-2
<400> 228
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Thr Tyr Gln Gly Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr
65 70 75 80
Met Glu Leu Arg Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Thr Arg Gly Ala Trp Phe Gly Glu Ser Leu Ile Gly
100 105 110
Gly Phe Asp Asn Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Ile
130 135 140
Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly Glu Arg
145 150 155 160
Ala Thr Leu Ser Cys Arg Ala Ser Gln Thr Val Ser Ser Thr Ser Leu
165 170 175
Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr
180 185 190
Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser Gly Ser
195 200 205
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu Pro Glu
210 215 220
Asp Phe Ala Val Tyr Tyr Cys Gln Gln His Asp Thr Ser Leu Thr Phe
225 230 235 240
Gly Gly Gly Thr Lys Val Glu Ile Lys
245
<210> 229
<211> 249
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность S309-scFv-(VL-VH)
v1.1 SARS-CoV-2
<400> 229
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Thr Val Ser Ser Thr
20 25 30
Ser Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln His Asp Thr Ser Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Gly Gly Gly Gly Ser
100 105 110
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln Leu Val Gln
115 120 125
Ser Gly Ala Glu Val Lys Lys Pro Gly Ala Ser Val Lys Val Ser Cys
130 135 140
Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr Gly Ile Ser Trp Val Arg
145 150 155 160
Gln Ala Pro Gly Gln Gly Leu Glu Trp Met Gly Trp Ile Ser Thr Tyr
165 170 175
Gln Gly Asn Thr Asn Tyr Ala Gln Lys Phe Gln Gly Arg Val Thr Met
180 185 190
Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr Met Glu Leu Arg Arg Leu
195 200 205
Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys Ala Arg Asp Tyr Thr Arg
210 215 220
Gly Ala Trp Phe Gly Glu Ser Leu Ile Gly Gly Phe Asp Asn Trp Gly
225 230 235 240
Gln Gly Thr Leu Val Thr Val Ser Ser
245
<210> 230
<211> 518
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
S309-scFv-(VH-VL)-(VH-VL) v1.1 SARS-CoV-2
<400> 230
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Thr Tyr Gln Gly Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr
65 70 75 80
Met Glu Leu Arg Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Thr Arg Gly Ala Trp Phe Gly Glu Ser Leu Ile Gly
100 105 110
Gly Phe Asp Asn Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Ile
130 135 140
Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly Glu Arg
145 150 155 160
Ala Thr Leu Ser Cys Arg Ala Ser Gln Thr Val Ser Ser Thr Ser Leu
165 170 175
Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr
180 185 190
Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser Gly Ser
195 200 205
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu Pro Glu
210 215 220
Asp Phe Ala Val Tyr Tyr Cys Gln Gln His Asp Thr Ser Leu Thr Phe
225 230 235 240
Gly Gly Gly Thr Lys Val Glu Ile Lys Gly Gly Gly Gly Ser Gly Gly
245 250 255
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln
260 265 270
Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala Ser Val Lys
275 280 285
Val Ser Cys Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr Gly Ile Ser
290 295 300
Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met Gly Trp Ile
305 310 315 320
Ser Thr Tyr Gln Gly Asn Thr Asn Tyr Ala Gln Lys Phe Gln Gly Arg
325 330 335
Val Thr Met Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr Met Glu Leu
340 345 350
Arg Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys Ala Arg Asp
355 360 365
Tyr Thr Arg Gly Ala Trp Phe Gly Glu Ser Leu Ile Gly Gly Phe Asp
370 375 380
Asn Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
385 390 395 400
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Ile Val Leu Thr
405 410 415
Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly Glu Arg Ala Thr Leu
420 425 430
Ser Cys Arg Ala Ser Gln Thr Val Ser Ser Thr Ser Leu Ala Trp Tyr
435 440 445
Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr Gly Ala Ser
450 455 460
Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly
465 470 475 480
Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu Pro Glu Asp Phe Ala
485 490 495
Val Tyr Tyr Cys Gln Gln His Asp Thr Ser Leu Thr Phe Gly Gly Gly
500 505 510
Thr Lys Val Glu Ile Lys
515
<210> 231
<211> 518
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
S309-scFv-(VH-VL)-(VL-VH) v1.1 SARS-CoV-2
<400> 231
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Thr Tyr Gln Gly Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr
65 70 75 80
Met Glu Leu Arg Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Thr Arg Gly Ala Trp Phe Gly Glu Ser Leu Ile Gly
100 105 110
Gly Phe Asp Asn Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Ile
130 135 140
Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly Glu Arg
145 150 155 160
Ala Thr Leu Ser Cys Arg Ala Ser Gln Thr Val Ser Ser Thr Ser Leu
165 170 175
Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr
180 185 190
Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser Gly Ser
195 200 205
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu Pro Glu
210 215 220
Asp Phe Ala Val Tyr Tyr Cys Gln Gln His Asp Thr Ser Leu Thr Phe
225 230 235 240
Gly Gly Gly Thr Lys Val Glu Ile Lys Gly Gly Gly Gly Ser Gly Gly
245 250 255
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Ile Val
260 265 270
Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly Glu Arg Ala
275 280 285
Thr Leu Ser Cys Arg Ala Ser Gln Thr Val Ser Ser Thr Ser Leu Ala
290 295 300
Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr Gly
305 310 315 320
Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser Gly Ser Gly
325 330 335
Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu Pro Glu Asp
340 345 350
Phe Ala Val Tyr Tyr Cys Gln Gln His Asp Thr Ser Leu Thr Phe Gly
355 360 365
Gly Gly Thr Lys Val Glu Ile Lys Gly Gly Gly Gly Ser Gly Gly Gly
370 375 380
Gly Ser Gly Gly Gly Gly Ser Gln Val Gln Leu Val Gln Ser Gly Ala
385 390 395 400
Glu Val Lys Lys Pro Gly Ala Ser Val Lys Val Ser Cys Lys Ala Ser
405 410 415
Gly Tyr Pro Phe Thr Ser Tyr Gly Ile Ser Trp Val Arg Gln Ala Pro
420 425 430
Gly Gln Gly Leu Glu Trp Met Gly Trp Ile Ser Thr Tyr Gln Gly Asn
435 440 445
Thr Asn Tyr Ala Gln Lys Phe Gln Gly Arg Val Thr Met Thr Thr Asp
450 455 460
Thr Ser Thr Thr Thr Gly Tyr Met Glu Leu Arg Arg Leu Arg Ser Asp
465 470 475 480
Asp Thr Ala Val Tyr Tyr Cys Ala Arg Asp Tyr Thr Arg Gly Ala Trp
485 490 495
Phe Gly Glu Ser Leu Ile Gly Gly Phe Asp Asn Trp Gly Gln Gly Thr
500 505 510
Leu Val Thr Val Ser Ser
515
<210> 232
<211> 518
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
S309-scFv-(VH-VL)-(VL-VH) v1.1 SARS-CoV-2
<400> 232
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Thr Val Ser Ser Thr
20 25 30
Ser Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln His Asp Thr Ser Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Gly Gly Gly Gly Ser
100 105 110
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln Leu Val Gln
115 120 125
Ser Gly Ala Glu Val Lys Lys Pro Gly Ala Ser Val Lys Val Ser Cys
130 135 140
Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr Gly Ile Ser Trp Val Arg
145 150 155 160
Gln Ala Pro Gly Gln Gly Leu Glu Trp Met Gly Trp Ile Ser Thr Tyr
165 170 175
Gln Gly Asn Thr Asn Tyr Ala Gln Lys Phe Gln Gly Arg Val Thr Met
180 185 190
Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr Met Glu Leu Arg Arg Leu
195 200 205
Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys Ala Arg Asp Tyr Thr Arg
210 215 220
Gly Ala Trp Phe Gly Glu Ser Leu Ile Gly Gly Phe Asp Asn Trp Gly
225 230 235 240
Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly
245 250 255
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln
260 265 270
Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala Ser Val Lys
275 280 285
Val Ser Cys Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr Gly Ile Ser
290 295 300
Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met Gly Trp Ile
305 310 315 320
Ser Thr Tyr Gln Gly Asn Thr Asn Tyr Ala Gln Lys Phe Gln Gly Arg
325 330 335
Val Thr Met Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr Met Glu Leu
340 345 350
Arg Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys Ala Arg Asp
355 360 365
Tyr Thr Arg Gly Ala Trp Phe Gly Glu Ser Leu Ile Gly Gly Phe Asp
370 375 380
Asn Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
385 390 395 400
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Ile Val Leu Thr
405 410 415
Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly Glu Arg Ala Thr Leu
420 425 430
Ser Cys Arg Ala Ser Gln Thr Val Ser Ser Thr Ser Leu Ala Trp Tyr
435 440 445
Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr Gly Ala Ser
450 455 460
Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly
465 470 475 480
Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu Pro Glu Asp Phe Ala
485 490 495
Val Tyr Tyr Cys Gln Gln His Asp Thr Ser Leu Thr Phe Gly Gly Gly
500 505 510
Thr Lys Val Glu Ile Lys
515
<210> 233
<211> 518
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
S309-scFv-(VL-VH)-(VL-VH) v1.1 SARS-CoV-2
<400> 233
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Thr Val Ser Ser Thr
20 25 30
Ser Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln His Asp Thr Ser Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Gly Gly Gly Gly Ser
100 105 110
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln Leu Val Gln
115 120 125
Ser Gly Ala Glu Val Lys Lys Pro Gly Ala Ser Val Lys Val Ser Cys
130 135 140
Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr Gly Ile Ser Trp Val Arg
145 150 155 160
Gln Ala Pro Gly Gln Gly Leu Glu Trp Met Gly Trp Ile Ser Thr Tyr
165 170 175
Gln Gly Asn Thr Asn Tyr Ala Gln Lys Phe Gln Gly Arg Val Thr Met
180 185 190
Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr Met Glu Leu Arg Arg Leu
195 200 205
Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys Ala Arg Asp Tyr Thr Arg
210 215 220
Gly Ala Trp Phe Gly Glu Ser Leu Ile Gly Gly Phe Asp Asn Trp Gly
225 230 235 240
Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly
245 250 255
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Ile Val
260 265 270
Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly Glu Arg Ala
275 280 285
Thr Leu Ser Cys Arg Ala Ser Gln Thr Val Ser Ser Thr Ser Leu Ala
290 295 300
Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr Gly
305 310 315 320
Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser Gly Ser Gly
325 330 335
Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu Pro Glu Asp
340 345 350
Phe Ala Val Tyr Tyr Cys Gln Gln His Asp Thr Ser Leu Thr Phe Gly
355 360 365
Gly Gly Thr Lys Val Glu Ile Lys Gly Gly Gly Gly Ser Gly Gly Gly
370 375 380
Gly Ser Gly Gly Gly Gly Ser Gln Val Gln Leu Val Gln Ser Gly Ala
385 390 395 400
Glu Val Lys Lys Pro Gly Ala Ser Val Lys Val Ser Cys Lys Ala Ser
405 410 415
Gly Tyr Pro Phe Thr Ser Tyr Gly Ile Ser Trp Val Arg Gln Ala Pro
420 425 430
Gly Gln Gly Leu Glu Trp Met Gly Trp Ile Ser Thr Tyr Gln Gly Asn
435 440 445
Thr Asn Tyr Ala Gln Lys Phe Gln Gly Arg Val Thr Met Thr Thr Asp
450 455 460
Thr Ser Thr Thr Thr Gly Tyr Met Glu Leu Arg Arg Leu Arg Ser Asp
465 470 475 480
Asp Thr Ala Val Tyr Tyr Cys Ala Arg Asp Tyr Thr Arg Gly Ala Trp
485 490 495
Phe Gly Glu Ser Leu Ile Gly Gly Phe Asp Asn Trp Gly Gln Gly Thr
500 505 510
Leu Val Thr Val Ser Ser
515
<210> 234
<211> 114
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VL (VK) S300-v14 mAb SARS-CoV-2
<400> 234
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Glu Arg Ala Thr Ile Asn Cys Lys Ser Ser Gln Ser Val Leu Tyr Ser
20 25 30
Ser Asn Asn Lys Asn Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Pro Pro Lys Leu Leu Ile Ser Trp Ala Ser Thr Arg Glu Ser Gly Val
50 55 60
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
65 70 75 80
Ile Ser Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Tyr Cys Gln Gln
85 90 95
Tyr Tyr Ser Ala Pro Gly Ile Thr Phe Gly Gln Gly Thr Arg Leu Glu
100 105 110
Ile Lys
<210> 235
<211> 12
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRL1 S300-v14 mAb SARS-CoV-2
<400> 235
Gln Ser Val Leu Tyr Ser Ser Asn Asn Lys Asn Tyr
1 5 10
<210> 236
<211> 3
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность SARS-CoV-2 S300-v14 mAb CDRL2
<400> 236
Trp Ala Ser
1
<210> 237
<211> 10
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRL3 S300-v14 mAb SARS-CoV-2
<400> 237
Gln Gln Tyr Tyr Ser Ala Pro Gly Ile Thr
1 5 10
<210> 238
<211> 343
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VL (VK) S300-v14 mAb SARS-CoV2
<400> 238
gacatcgtga tgacccagtc tccagactca ctggctgtgt ctctgggcga gagggccacc 60
atcaactgta agtccagcca gagtgtttta tacagctcca acaataagaa ctacttagct 120
tggtaccagc agaaaccagg acagcctcct aagctgctca tttcctgggc ttctacccgg 180
gaatccgggg tccctgaccg attcagtggc agcgggtctg ggacagattt cactctcacc 240
atcagcagcc tgcaggctga agatgtggca gtttattact gtcaacaata ttatagtgct 300
cccgggatca ccttcggcca ggggacacga ctggagatta aac 343
<210> 239
<211> 129
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S307 mAb SARS-CoV2
<400> 239
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Val Thr Ser Gly
20 25 30
Ser Tyr Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Tyr Met Tyr Tyr Ser Gly Ser Thr Asn Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Ala Gly Cys Thr Gly Ile Thr Cys Leu Arg Tyr Asp Tyr
100 105 110
Tyr Tyr Gly Leu Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser
115 120 125
Ser
<210> 240
<211> 10
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH1 S307 mAb SARS-CoV2
<400> 240
Gly Gly Ser Val Thr Ser Gly Ser Tyr Tyr
1 5 10
<210> 241
<211> 7
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH2 S307 mAb SARS-CoV2
<400> 241
Met Tyr Tyr Ser Gly Ser Thr
1 5
<210> 242
<211> 21
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRH3 S307 mAb SARS-CoV2
<400> 242
Ala Arg Ala Gly Cys Thr Gly Ile Thr Cys Leu Arg Tyr Asp Tyr Tyr
1 5 10 15
Tyr Gly Leu Asp Val
20
<210> 243
<211> 108
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VL(VK) S307 mAb SARS-CoV2
<400> 243
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Lys Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Arg Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Ser Arg Ala Ala Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Ser Ser Ser
85 90 95
Trp Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 244
<211> 7
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRL1 S307 mAb SARS-CoV2
<400> 244
Gln Ser Val Ser Ser Ser Tyr
1 5
<210> 245
<211> 3
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRL2 S307 mAb SARS-CoV2
<400> 245
Gly Ala Ser
1
<210> 246
<211> 9
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CDRL3 S307 mAb SARS-CoV2
<400> 246
Gln Gln Tyr Gly Ser Ser Ser Trp Thr
1 5
<210> 247
<211> 387
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S307 mAb SARS-CoV2
<400> 247
caggtgcagc tgcaggagtc gggcccagga ctggtgaagc cttcggagac cctgtccctc 60
acctgcactg tctctggtgg ctccgtcacc agtggtagtt actactggag ctggatccgg 120
cagcccccag ggaagggact ggagtggatt gggtatatgt attacagtgg gagcaccaat 180
tacaacccct ccctcaagag tcgagtcacc atatcagtag acacgtccaa gaaccagttc 240
tccctgaagc tgagctctgt gaccgctgcg gacacggccg tgtattactg tgcgagggca 300
ggttgtactg gtatcacctg cttacgttac gactactact acggtctgga cgtctggggc 360
caagggacca cggtcaccgt ctcctca 387
<210> 248
<211> 325
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VL(VK) S307 mAb SARS-CoV2
<400> 248
gaaattgtgt tgacgcagtc tccaggcacc ctgtctttgt ctccagggaa aagagccacc 60
ctctcctgca gggccagtca gagtgttagc agcagctact tagcctggta ccagcagaga 120
cctggccagg ctcccaggct cctcatctat ggtgcatcca gcagggccgc tggcatccca 180
gacaggttca gtggcagtgg gtctgggaca gacttcactc tcaccatcag cagactggag 240
cctgaagatt ttgcagtgta ttactgtcag cagtatggta gctcatcgtg gacgttcggc 300
caagggacca aggtggaaat caaac 325
<210> 249
<211> 381
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VH S309-v1 mAb SARS-CoV-2
<400> 249
caggttcagc tggtgcagtc tggagctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggtta cccctttacc agttatggta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggatgg atcagcactt acaatggtaa cacaaattat 180
gcacagaagt tccagggcag agtcaccatg accacagaca catccacgac cacaggctac 240
atggagctga ggaggctgag atctgacgac acggccgtgt attactgtgc gagagattat 300
actcgtggtg cttggttcgg ggagtcattg atagggggct ttgacaactg gggccaggga 360
accctggtca ccgtctcctc a 381
<210> 250
<211> 322
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность VL(VK) S309-v13 mAb SAES-CoV-2
<400> 250
gaaattgtgt tgacgcagtc tccaggcacc ctgtctttgt ctccagggga aagagccacc 60
ctctcctgca gggccagtca gactgttagc agcacctcct tagcctggta ccagcagaaa 120
cctggccagg ctcccaggct cctcatctat ggtgcatcca gcagggccac tggcatccca 180
gacaggttca gcggcagtgg gtctgggaca gacttcactc tcaccatcag cagactggag 240
cctgaagatt ttgcagtgta ttactgtcag cagcatgata cctcactcac tttcggcgga 300
gggaccaagg tggagatcaa ac 322
<210> 251
<211> 767
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность промотер CMV
<400> 251
gacattgatt attgactagt tattaatagt aatcaattac ggggtcatta gttcatagcc 60
catatatgga gttccgcgtt acataactta cggtaaatgg cccgcctggc tgaccgccca 120
acgacccccg cccatgacgt caataatgac gtatgttccc atagtaacgc caatagggac 180
tttccattga cgtcaatggg tggagtattt acggtaaact gcccacttgg cagtacatca 240
agtgtatcat atgccaagta cgccccctat tgacgtcaat gacggtaaat ggcccgcctg 300
gcattatgcc cagtacatga ccttatggga ctttcctact tggcagtaca tctacgtatt 360
agtcatcgct attaccatgg tgatgcggtt ttggcagtac atcaatgggc gtggatagcg 420
gtttgactca cggggatttc caagtctcca ccccattgac gtcaatggga gtttgttttg 480
gcaccaaaat caacgggact ttccaaaatg tcgtaacaac tccgccccat tgacgcaaat 540
gggcggtagg cgtgtacggt gggaggtcta tataagcaga gctcgtttag tgaaccgtca 600
gatcgcctgg agacgccatc cacgctgttt tgacctccat agaagacacc gggaccgatc 660
cagcctccgc ggccgggaac ggtgcattgg aacgcggatt ccccgtgcca agagtgacgt 720
aagtaccgcc tatagagtct ataggcccac ccccttggct tcgttag 767
<210> 252
<211> 50
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Сигнальный пептид
<400> 252
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggtgt 50
<210> 253
<211> 135
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Сигнальная последовательность полиаденилирования
<400> 253
aacttgttta ttgcagctta taatggttac aaataaagca atagcatcac aaatttcaca 60
aataaagcat ttttttcact gcattctagt tgtggtttgt ccaaactcat caatgtatct 120
tatcatgtct ggatc 135
<210> 254
<211> 323
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
легкой цепи IgKC*01 SARS-CoV-2
<400> 254
gtacggtggc tgcaccatct gtcttcatct tcccgccatc tgatgagcag ttgaaatctg 60
gaactgcctc tgttgtgtgc ctgctgaata acttctatcc cagagaggcc aaagtacagt 120
ggaaggtgga taacgccctc caatcgggta actcccagga gagtgtcaca gagcaggaca 180
gcaaggacag cacctacagc ctcagcagca ccctgacgct gagcaaagca gactacgaga 240
aacacaaagt ctacgcctgc gaagtcaccc atcagggcct gagctcgccc gtcacaaaga 300
gcttcaacag gggagagtgt tag 323
<210> 255
<211> 993
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CH1-CH3 G1m17;
IgG1*01 SARS-CoV-2
<400> 255
gcgtcgacca agggcccatc ggtcttcccc ctggcaccct cctccaagag cacctctggg 60
ggcacagcgg ccctgggctg cctggtcaag gactacttcc ccgaacctgt gacggtctcg 120
tggaactcag gcgccctgac cagcggcgtg cacaccttcc cggctgtcct acagtcctca 180
ggactctact ccctcagcag cgtggtgacc gtgccctcca gcagcttggg cacccagacc 240
tacatctgca acgtgaatca caagcccagc aacaccaagg tggacaagag agttgagccc 300
aaatcttgtg acaaaactca cacatgccca ccgtgcccag cacctgaact cctgggggga 360
ccgtcagtct tcctcttccc cccaaaaccc aaggacaccc tcatgatctc ccggacccct 420
gaggtcacat gcgtggtggt ggacgtgagc cacgaagacc ctgaggtcaa gttcaactgg 480
tacgtggacg gcgtggaggt gcataatgcc aagacaaagc cgcgggagga gcagtacaac 540
agcacgtacc gtgtggtcag cgtcctcacc gtcctgcacc aggactggct gaatggcaag 600
gagtacaagt gcaaggtctc caacaaagcc ctcccagccc ccatcgagaa aaccatctcc 660
aaagccaaag ggcagccccg agaaccacag gtgtacaccc tgcccccatc ccgggaggag 720
atgaccaaga accaggtcag cctgacctgc ctggtcaaag gcttctatcc cagcgacatc 780
gccgtggagt gggagagcaa tgggcagccg gagaacaact acaagaccac gcctcccgtg 840
ctggactccg acggctcctt cttcctctat agcaagctca ccgtggacaa gagcaggtgg 900
cagcagggga acgtcttctc atgctccgtg atgcatgagg ctctgcacaa ccactacacg 960
cagaagagcc tctccctgtc cccgggtaaa tga 993
<210> 256
<211> 16
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Сигнальный пептид
<400> 256
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
<210> 257
<211> 1431
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CH1-CH3 S309-v1.1 mAb
G1m17 SARS-CoV-2; IgGHG1*01 LS; GAALIE; сигнальный
пептид (nt-CO)
<400> 257
atgggctggt cctgtatcat cctgttcctg gtcgccacag ccaccggagt gcacagccaa 60
gtgcagctgg tccagagcgg cgccgaggtg aagaagcccg gcgctagcgt gaaggtgtcc 120
tgtaaagcca gcggatatcc ttttaccagc tacggcatct cctgggtgcg gcaggcccct 180
ggccagggcc tggaatggat gggctggatc agcacctacc agggaaatac caactacgcc 240
cagaagttcc agggaagagt gacaatgacc acagatacat ctacaaccac cggctacatg 300
gaactgaggc ggctgagaag cgacgacacc gccgtgtact actgcgccag agattacacc 360
agaggcgctt ggttcggcga gagcctgatc ggcggcttcg acaactgggg ccagggaacc 420
ctggtgacag tgtctagcgc ttctaccaaa ggcccttctg tctttcctct ggccccttct 480
agcaagtcta caagcggagg caccgccgcc ctgggctgcc tggtgaagga ctacttcccc 540
gagcccgtga ccgtgagctg gaatagcggc gccctgacaa gcggcgtgca caccttccca 600
gctgtgctgc agagcagcgg cctgtatagc ctgagcagcg tggtcaccgt gcccagcagc 660
agcctgggaa cacagaccta catctgcaac gtgaaccaca agccttctaa taccaaggtg 720
gataagaagg tggaacctaa gagctgcgac aaaacacaca catgccctcc atgtcctgct 780
ccagagctgc tggccggccc cagcgttttt ctgttccccc ccaaacctaa agacaccctg 840
atgatcagca gaacccctga ggtgacctgt gtggtggtgg acgtgtccca cgaagatcct 900
gaggtgaagt tcaactggta cgtggatgga gtggaagtgc acaacgccaa gaccaaacct 960
agagaagagc agtacaacag cacatataga gtcgtgtccg tgcttacagt gctgcaccag 1020
gactggctga atggaaagga atacaagtgc aaggtgtcca acaaggccct gcctctgcct 1080
gaggagaaga caatctctaa agccaagggc caacctcggg aacctcaggt gtacacactg 1140
ccccccagcc gggacgagct gaccaagaac caggtgtccc tgacctgcct ggtcaagggc 1200
ttctacccct ctgatatcgc cgtggaatgg gagagcaacg gccaacctga gaacaactac 1260
aagaccaccc ctccagtgct ggacagcgac ggcagcttct tcctgtacag caagctgacc 1320
gttgacaagt ccagatggca gcagggcaac gtgttcagct gtagcgtcct gcacgaggcc 1380
ctgcattctc actacaccca gaagagcctg tccctcagcc ctggcaagtg a 1431
<210> 258
<211> 1374
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CH1-CH3 S309-v1.1 mAb
G1m17 SARS-CoV-2; IgGHG1*01 LS; GAALIE; (nt-CO)
<400> 258
caagtgcagc tggtccagag cggcgccgag gtgaagaagc ccggcgctag cgtgaaggtg 60
tcctgtaaag ccagcggata tccttttacc agctacggca tctcctgggt gcggcaggcc 120
cctggccagg gcctggaatg gatgggctgg atcagcacct accagggaaa taccaactac 180
gcccagaagt tccagggaag agtgacaatg accacagata catctacaac caccggctac 240
atggaactga ggcggctgag aagcgacgac accgccgtgt actactgcgc cagagattac 300
accagaggcg cttggttcgg cgagagcctg atcggcggct tcgacaactg gggccaggga 360
accctggtga cagtgtctag cgcttctacc aaaggccctt ctgtctttcc tctggcccct 420
tctagcaagt ctacaagcgg aggcaccgcc gccctgggct gcctggtgaa ggactacttc 480
cccgagcccg tgaccgtgag ctggaatagc ggcgccctga caagcggcgt gcacaccttc 540
ccagctgtgc tgcagagcag cggcctgtat agcctgagca gcgtggtcac cgtgcccagc 600
agcagcctgg gaacacagac ctacatctgc aacgtgaacc acaagccttc taataccaag 660
gtggataaga aggtggaacc taagagctgc gacaaaacac acacatgccc tccatgtcct 720
gctccagagc tgctggccgg ccccagcgtt tttctgttcc cccccaaacc taaagacacc 780
ctgatgatca gcagaacccc tgaggtgacc tgtgtggtgg tggacgtgtc ccacgaagat 840
cctgaggtga agttcaactg gtacgtggat ggagtggaag tgcacaacgc caagaccaaa 900
cctagagaag agcagtacaa cagcacatat agagtcgtgt ccgtgcttac agtgctgcac 960
caggactggc tgaatggaaa ggaatacaag tgcaaggtgt ccaacaaggc cctgcctctg 1020
cctgaggaga agacaatctc taaagccaag ggccaacctc gggaacctca ggtgtacaca 1080
ctgcccccca gccgggacga gctgaccaag aaccaggtgt ccctgacctg cctggtcaag 1140
ggcttctacc cctctgatat cgccgtggaa tgggagagca acggccaacc tgagaacaac 1200
tacaagacca cccctccagt gctggacagc gacggcagct tcttcctgta cagcaagctg 1260
accgttgaca agtccagatg gcagcagggc aacgtgttca gctgtagcgt cctgcacgag 1320
gccctgcatt ctcactacac ccagaagagc ctgtccctca gccctggcaa gtga 1374
<210> 259
<211> 1431
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CH1-CH3 S309-v1.1 mAb
G1m17 SARS-CoV-2; IgGHG1*01 LS; сигнальный пептид
(nt-CO)
<400> 259
atgggctggt cctgtatcat cctgttcctg gtcgccacag ccaccggagt gcacagccaa 60
gtgcagctgg tccagagcgg cgccgaggtg aagaagcccg gcgctagcgt gaaggtgtcc 120
tgtaaagcca gcggatatcc ttttaccagc tacggcatct cctgggtgcg gcaggcccct 180
ggccagggcc tggaatggat gggctggatc agcacctacc agggaaatac caactacgcc 240
cagaagttcc agggaagagt gacaatgacc acagatacat ctacaaccac cggctacatg 300
gaactgaggc ggctgagaag cgacgacacc gccgtgtact actgcgccag agattacacc 360
agaggcgctt ggttcggcga gagcctgatc ggcggcttcg acaactgggg ccagggaacc 420
ctggtgacag tgtctagcgc ttctaccaaa ggcccttctg tctttcctct ggccccttct 480
agcaagtcta caagcggagg caccgccgcc ctgggctgcc tggtgaagga ctacttcccc 540
gagcccgtga ccgtgagctg gaatagcggc gccctgacaa gcggcgtgca caccttccca 600
gctgtgctgc agagcagcgg cctgtatagc ctgagcagcg tggtcaccgt gcccagcagc 660
agcctgggaa cacagaccta catctgcaac gtgaaccaca agccttctaa taccaaggtg 720
gataagaagg tggaacctaa gagctgcgac aaaacacaca catgccctcc atgtcctgct 780
ccagagctgc tgggcggccc cagcgttttt ctgttccccc ccaaacctaa agacaccctg 840
atgatcagca gaacccctga ggtgacctgt gtggtggtgg acgtgtccca cgaagatcct 900
gaggtgaagt tcaactggta cgtggatgga gtggaagtgc acaacgccaa gaccaaacct 960
agagaagagc agtacaacag cacatataga gtcgtgtccg tgcttacagt gctgcaccag 1020
gactggctga atggaaagga atacaagtgc aaggtgtcca acaaggccct gcctgcccct 1080
atcgagaaga caatctctaa agccaagggc caacctcggg aacctcaggt gtacacactg 1140
ccccccagcc gggacgagct gaccaagaac caggtgtccc tgacctgcct ggtcaagggc 1200
ttctacccct ctgatatcgc cgtggaatgg gagagcaacg gccaacctga gaacaactac 1260
aagaccaccc ctccagtgct ggacagcgac ggcagcttct tcctgtacag caagctgacc 1320
gttgacaagt ccagatggca gcagggcaac gtgttcagct gtagcgtcct gcacgaggcc 1380
ctgcattctc actacaccca gaagagcctg tccctcagcc ctggcaagtg a 1431
<210> 260
<211> 1374
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CH1-CH3 S309-v1.1 mAb
G1m17 SARS-CoV-2; IgGHG1*01 LS (nt-CO)
<400> 260
caagtgcagc tggtccagag cggcgccgag gtgaagaagc ccggcgctag cgtgaaggtg 60
tcctgtaaag ccagcggata tccttttacc agctacggca tctcctgggt gcggcaggcc 120
cctggccagg gcctggaatg gatgggctgg atcagcacct accagggaaa taccaactac 180
gcccagaagt tccagggaag agtgacaatg accacagata catctacaac caccggctac 240
atggaactga ggcggctgag aagcgacgac accgccgtgt actactgcgc cagagattac 300
accagaggcg cttggttcgg cgagagcctg atcggcggct tcgacaactg gggccaggga 360
accctggtga cagtgtctag cgcttctacc aaaggccctt ctgtctttcc tctggcccct 420
tctagcaagt ctacaagcgg aggcaccgcc gccctgggct gcctggtgaa ggactacttc 480
cccgagcccg tgaccgtgag ctggaatagc ggcgccctga caagcggcgt gcacaccttc 540
ccagctgtgc tgcagagcag cggcctgtat agcctgagca gcgtggtcac cgtgcccagc 600
agcagcctgg gaacacagac ctacatctgc aacgtgaacc acaagccttc taataccaag 660
gtggataaga aggtggaacc taagagctgc gacaaaacac acacatgccc tccatgtcct 720
gctccagagc tgctgggcgg ccccagcgtt tttctgttcc cccccaaacc taaagacacc 780
ctgatgatca gcagaacccc tgaggtgacc tgtgtggtgg tggacgtgtc ccacgaagat 840
cctgaggtga agttcaactg gtacgtggat ggagtggaag tgcacaacgc caagaccaaa 900
cctagagaag agcagtacaa cagcacatat agagtcgtgt ccgtgcttac agtgctgcac 960
caggactggc tgaatggaaa ggaatacaag tgcaaggtgt ccaacaaggc cctgcctgcc 1020
cctatcgaga agacaatctc taaagccaag ggccaacctc gggaacctca ggtgtacaca 1080
ctgcccccca gccgggacga gctgaccaag aaccaggtgt ccctgacctg cctggtcaag 1140
ggcttctacc cctctgatat cgccgtggaa tgggagagca acggccaacc tgagaacaac 1200
tacaagacca cccctccagt gctggacagc gacggcagct tcttcctgta cagcaagctg 1260
accgttgaca agtccagatg gcagcagggc aacgtgttca gctgtagcgt cctgcacgag 1320
gccctgcatt ctcactacac ccagaagagc ctgtccctca gccctggcaa gtga 1374
<210> 261
<211> 701
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность легкой цепи S309-v13 k1m3;
IgKC*01; сигнальный пептид (nt-CO)
<400> 261
tgggctggtc ctgcatcatc ctgttcctgg tggccacagc caccggcgtg cacagcgaga 60
tcgtcctgac acagagcccc ggcacactga gcctctcccc aggcgagcgg gctacactgt 120
cttgtagagc ttctcagacc gtgtccagca ccagcctggc ctggtatcag cagaaacctg 180
gccaggcccc tagactgctg atctacggcg ccagcagcag agccaccggc atccctgata 240
gattcagcgg cagcggatct ggaaccgact tcaccctgac catcagccgg ctggaacccg 300
aggactttgc cgtgtactac tgccagcaac acgacaccag cctgaccttc ggcggcggaa 360
caaaggtgga aatcaagaga accgtggccg cccctagcgt gttcatcttc ccccccagcg 420
acgagcagct gaagagcggt acagcttctg tggtgtgcct gctgaacaac ttctacccgc 480
gggaagccaa ggtgcagtgg aaggtggaca acgccctgca gagcggcaac agccaggaga 540
gcgtgacaga gcaggacagc aaggacagca cctacagcct gagcagcacc ctgaccctga 600
gcaaggccga ctacgagaag cacaaggtgt acgcctgtga agtgacccac cagggcctgt 660
ctagccctgt gaccaagtct tttaacagag gcgagtgctg a 701
<210> 262
<211> 645
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность легкой цепи S309-v13 k1m3;
IgKC*01; (nt-CO)
<400> 262
gagatcgtcc tgacacagag ccccggcaca ctgagcctct ccccaggcga gcgggctaca 60
ctgtcttgta gagcttctca gaccgtgtcc agcaccagcc tggcctggta tcagcagaaa 120
cctggccagg cccctagact gctgatctac ggcgccagca gcagagccac cggcatccct 180
gatagattca gcggcagcgg atctggaacc gacttcaccc tgaccatcag ccggctggaa 240
cccgaggact ttgccgtgta ctactgccag caacacgaca ccagcctgac cttcggcggc 300
ggaacaaagg tggaaatcaa gagaaccgtg gccgccccta gcgtgttcat cttccccccc 360
agcgacgagc agctgaagag cggtacagct tctgtggtgt gcctgctgaa caacttctac 420
ccgcgggaag ccaaggtgca gtggaaggtg gacaacgccc tgcagagcgg caacagccag 480
gagagcgtga cagagcagga cagcaaggac agcacctaca gcctgagcag caccctgacc 540
ctgagcaagg ccgactacga gaagcacaag gtgtacgcct gtgaagtgac ccaccagggc 600
ctgtctagcc ctgtgaccaa gtcttttaac agaggcgagt gctga 645
<210> 263
<211> 57
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Сигнальный пептид
<400> 263
atgggctggt cctgcatcat cctgttcctg gtggccacag ccaccggcgt gcacagc 57
<210> 264
<211> 19
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Сигнальный пептид
<400> 264
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser
<210> 265
<211> 329
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CH1-CH3 G1m17 SARS-CoV-2;
IgG1*01 LS без C-term Lys
<400> 265
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys
1 5 10 15
Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr
65 70 75 80
Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys
100 105 110
Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro
115 120 125
Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys
130 135 140
Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp
145 150 155 160
Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu
165 170 175
Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu
180 185 190
His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn
195 200 205
Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly
210 215 220
Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu
225 230 235 240
Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr
245 250 255
Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn
260 265 270
Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe
275 280 285
Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn
290 295 300
Val Phe Ser Cys Ser Val Leu His Glu Ala Leu His Ser His Tyr Thr
305 310 315 320
Gln Lys Ser Leu Ser Leu Ser Pro Gly
325
<210> 266
<211> 329
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность CH1-CH3 G1m17 SARS-CoV-2;
IgG1*01 LS GAALIE без C-term Lys
<400> 266
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys
1 5 10 15
Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr
65 70 75 80
Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys
100 105 110
Pro Ala Pro Glu Leu Leu Ala Gly Pro Ser Val Phe Leu Phe Pro Pro
115 120 125
Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys
130 135 140
Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp
145 150 155 160
Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu
165 170 175
Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu
180 185 190
His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn
195 200 205
Lys Ala Leu Pro Leu Pro Glu Glu Lys Thr Ile Ser Lys Ala Lys Gly
210 215 220
Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu
225 230 235 240
Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr
245 250 255
Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn
260 265 270
Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe
275 280 285
Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn
290 295 300
Val Phe Ser Cys Ser Val Leu His Glu Ala Leu His Ser His Tyr Thr
305 310 315 320
Gln Lys Ser Leu Ser Leu Ser Pro Gly
325
<210> 267
<211> 127
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Консенсусная последовательность VH S309 SARS-CoV-2
<220>
<221> ВАРИАНТ
<222> 50
<223> Xaa представляет собой W, F или Y
<220>
<221> ВАРИАНТ
<222> 55
<223> Xaa представляет собой N, Q, L или T
<220>
<221> ВАРИАНТ
<222> 56
<223> Xaa представляет собой G, S, A или Q
<220>
<221> ВАРИАНТ
<222> 105
<223> Xaa представляет собой W, F или Y
<400> 267
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Pro Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Xaa Ile Ser Thr Tyr Xaa Xaa Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Thr Thr Gly Tyr
65 70 75 80
Met Glu Leu Arg Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Thr Arg Gly Ala Xaa Phe Gly Glu Ser Leu Ile Gly
100 105 110
Gly Phe Asp Asn Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Однодоменное антитело и его модификации, специфически связывающиеся с RBD S белка вируса SARS-CoV-2, и способ их применения для терапии и экстренной профилактики заболеваний, вызываемых вирусом SARS-CoV-2 | 2021 |
|
RU2763001C1 |
| Вакцина на основе AAV5 для индукции специфического иммунитета к вирусу SARS-CoV-2 и/или профилактики коронавирусной инфекции, вызванной SARS-CoV-2 | 2020 |
|
RU2760301C1 |
| Вакцина на основе AAV5 для индукции специфического иммунитета к вирусу SARS-CoV-2 и/или профилактики коронавирусной инфекции, вызванной SARS-CoV-2 | 2021 |
|
RU2761879C1 |
| Вакцина на основе AAV5 для индукции специфического иммунитета к вирусу SARS-CoV-2 и/или профилактики коронавирусной инфекции, вызванной SARS-CoV-2 | 2020 |
|
RU2783313C1 |
| УСОВЕРШЕНСТВОВАННАЯ КОРОНАВИРУСНАЯ ВАКЦИНА | 2022 |
|
RU2816182C2 |
| БЕЛОК И ВАКЦИНА ПРОТИВ ИНФЕКЦИИ SARS-CoV-2 | 2020 |
|
RU2815060C1 |
| СПОСОБ ПОВЫШЕНИЯ ИММУНОГЕННОСТИ БЕЛКОВОГО/ПЕПТИДНОГО АНТИГЕНА | 2021 |
|
RU2830627C1 |
| Выделенный рекомбинантный вирус на основе вируса гриппа для индукции специфического иммунитета к вирусу гриппа и/или профилактики заболеваний, вызванных вирусом гриппа | 2021 |
|
RU2813150C2 |
| ВАКЦИНЫ НА ОСНОВЕ ИНФОРМАЦИОННОЙ РНК ПРОТИВ ШИРОКОГО СПЕКТРА ВАРИАНТОВ КОРОНАВИРУСА | 2022 |
|
RU2826172C2 |
| Генная конструкция для экспрессии рекомбинантных белков на основе участка S-белка SARS-CoV-2, включающего RBD и SD1, слитого с Fc фрагментом IgG, способ получения рекомбинантных белков, антигены и антигенные композиции для индукции длительного антительного и клеточного иммунитета против вируса SARS-CoV-2 | 2021 |
|
RU2802825C2 |
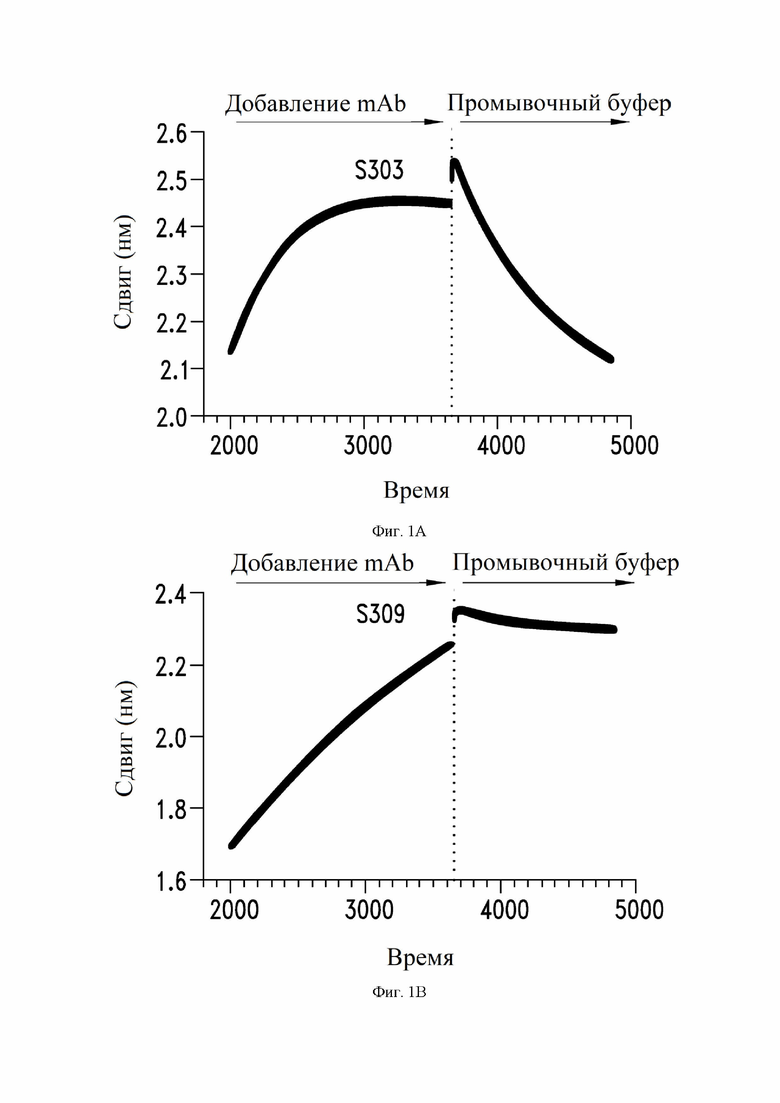

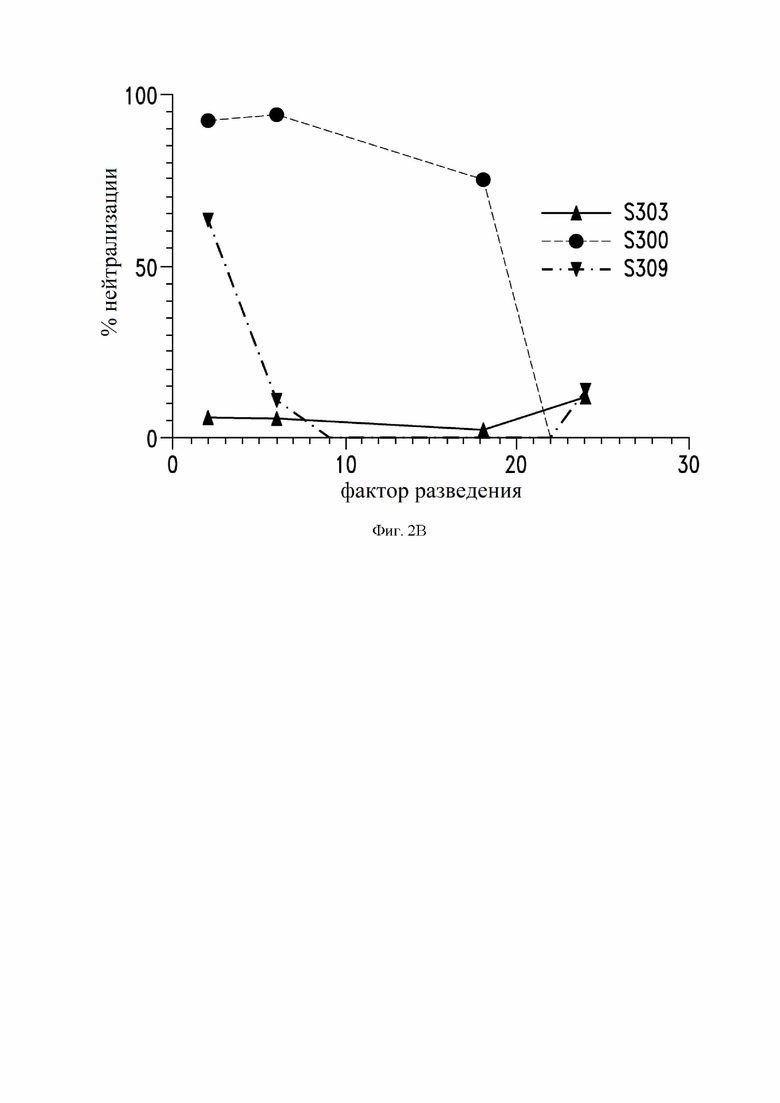
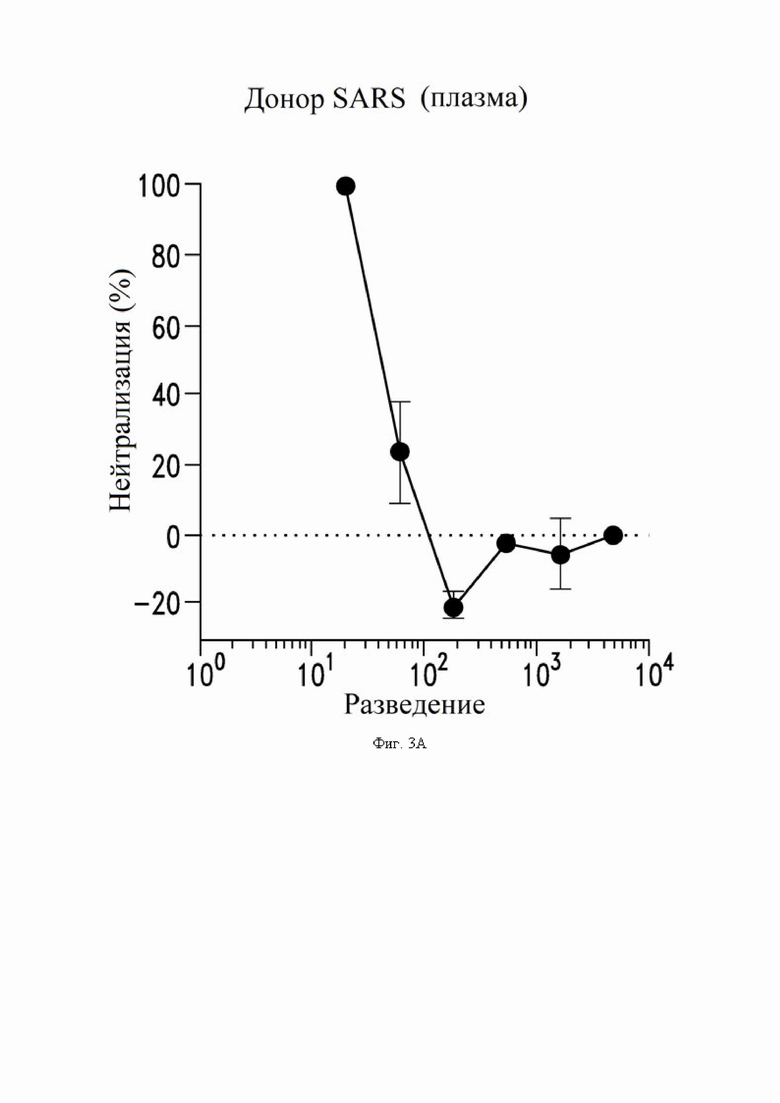
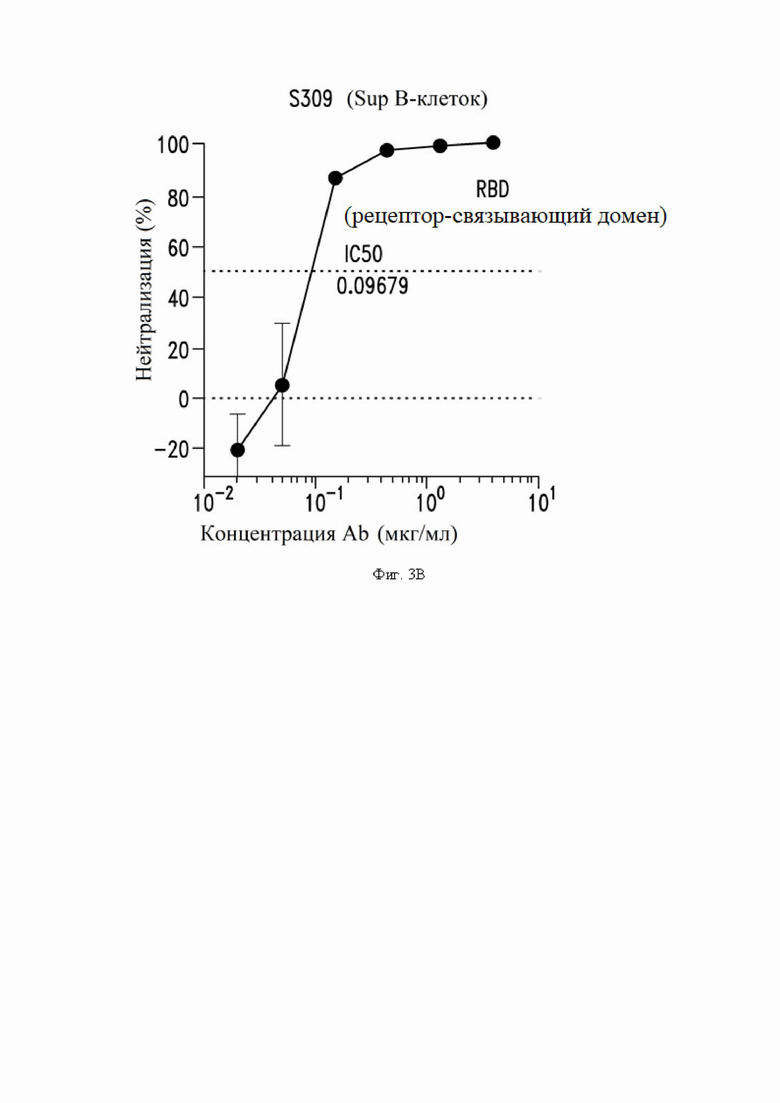
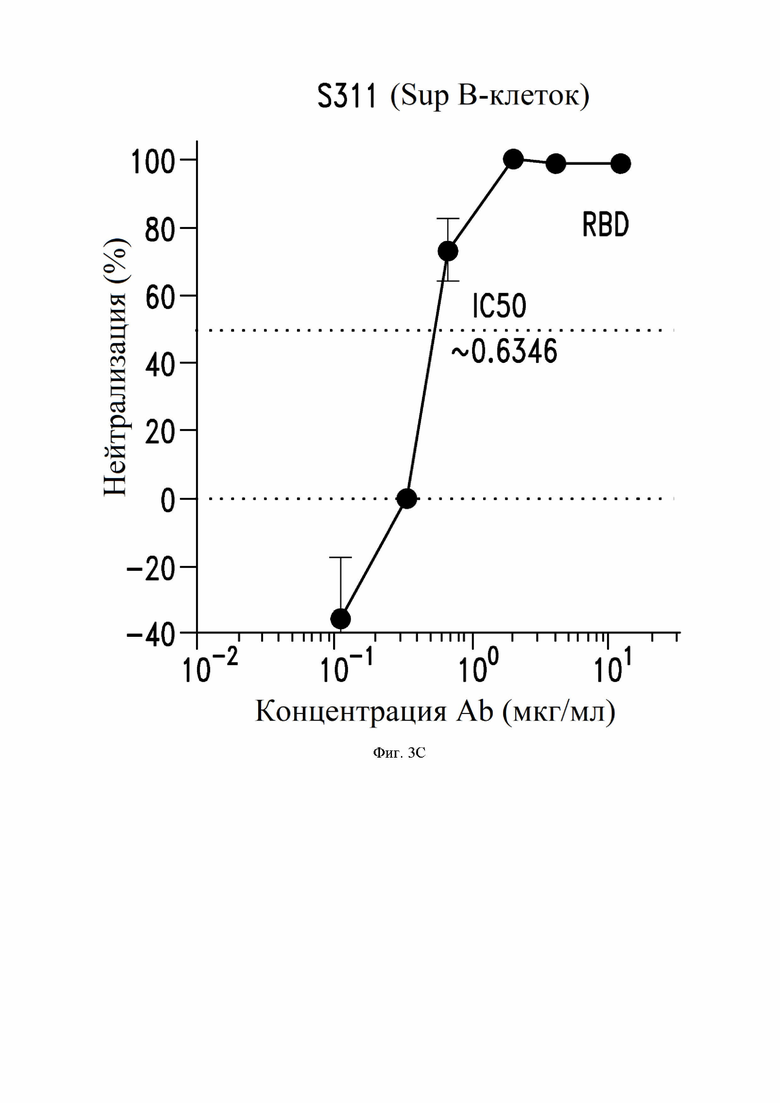
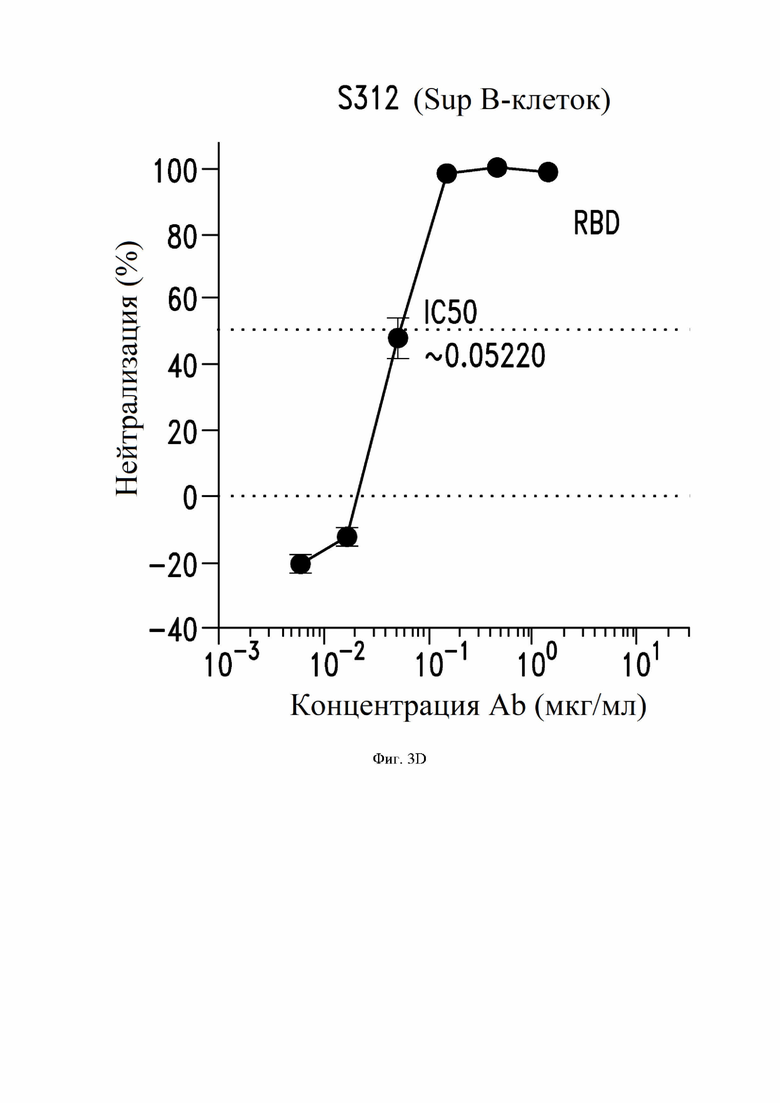
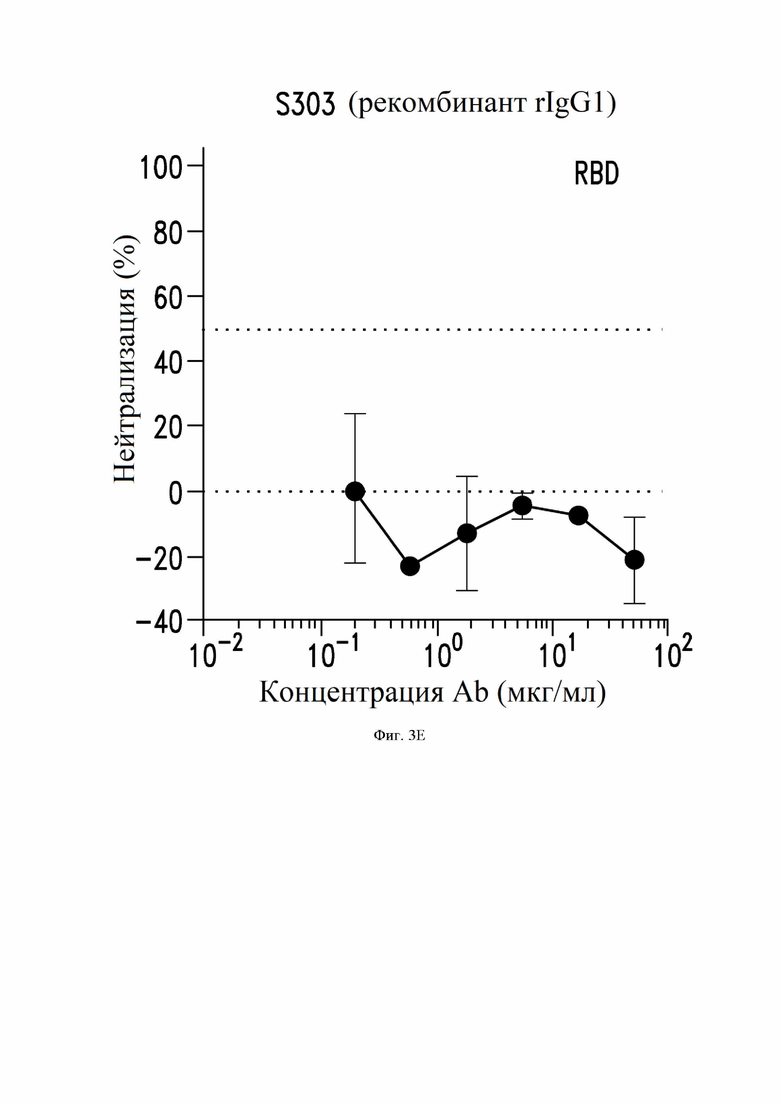
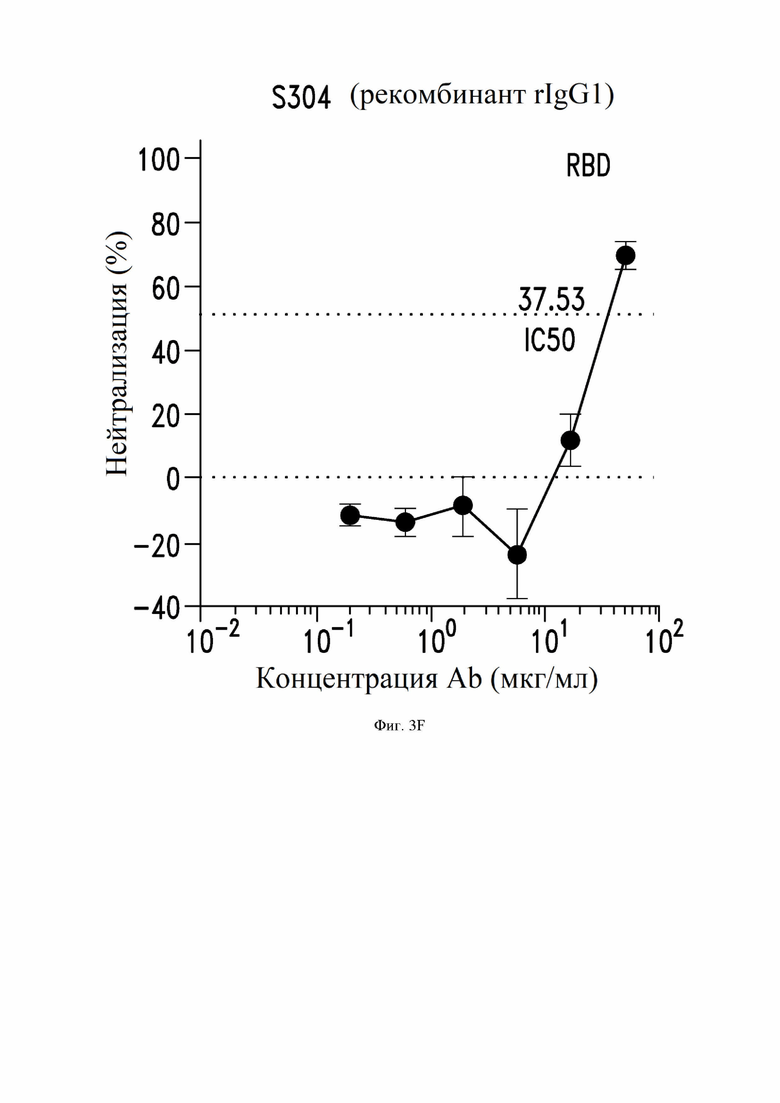
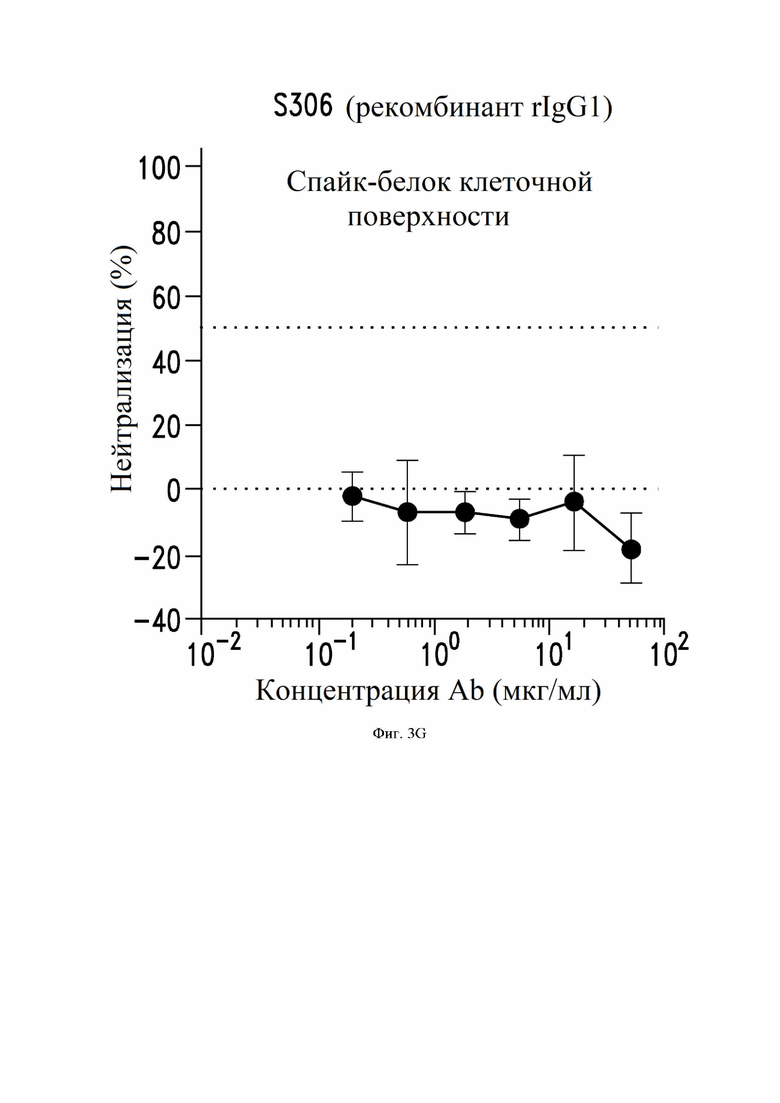
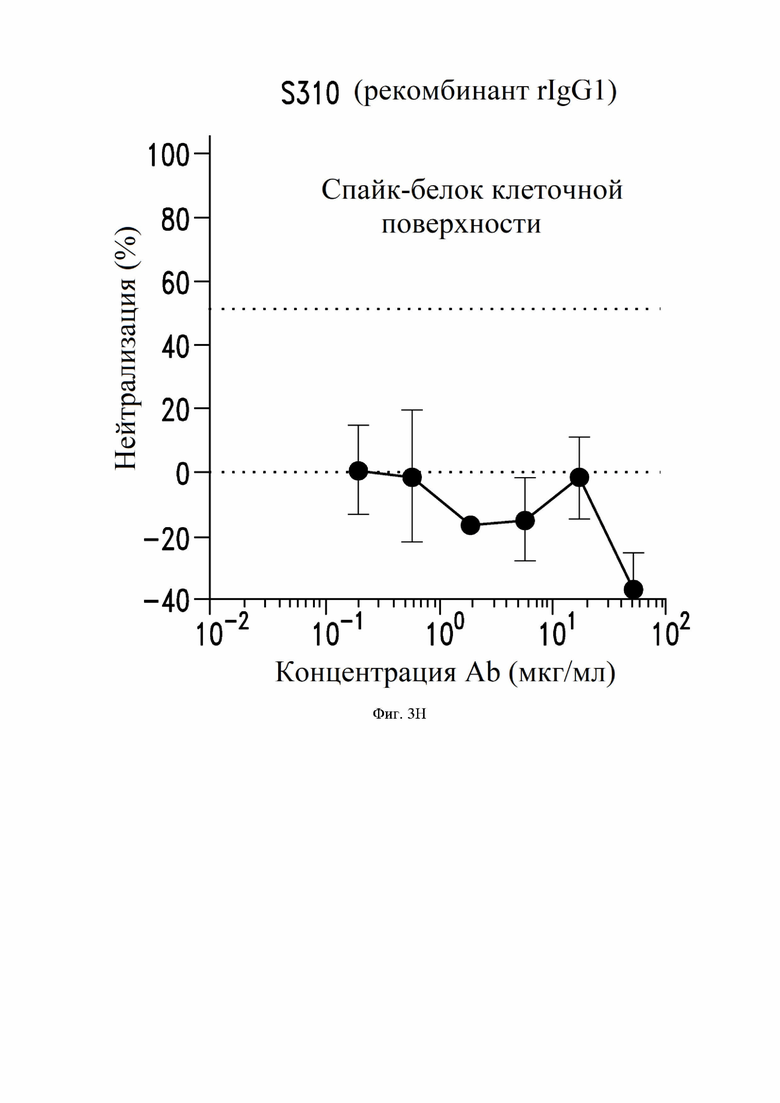
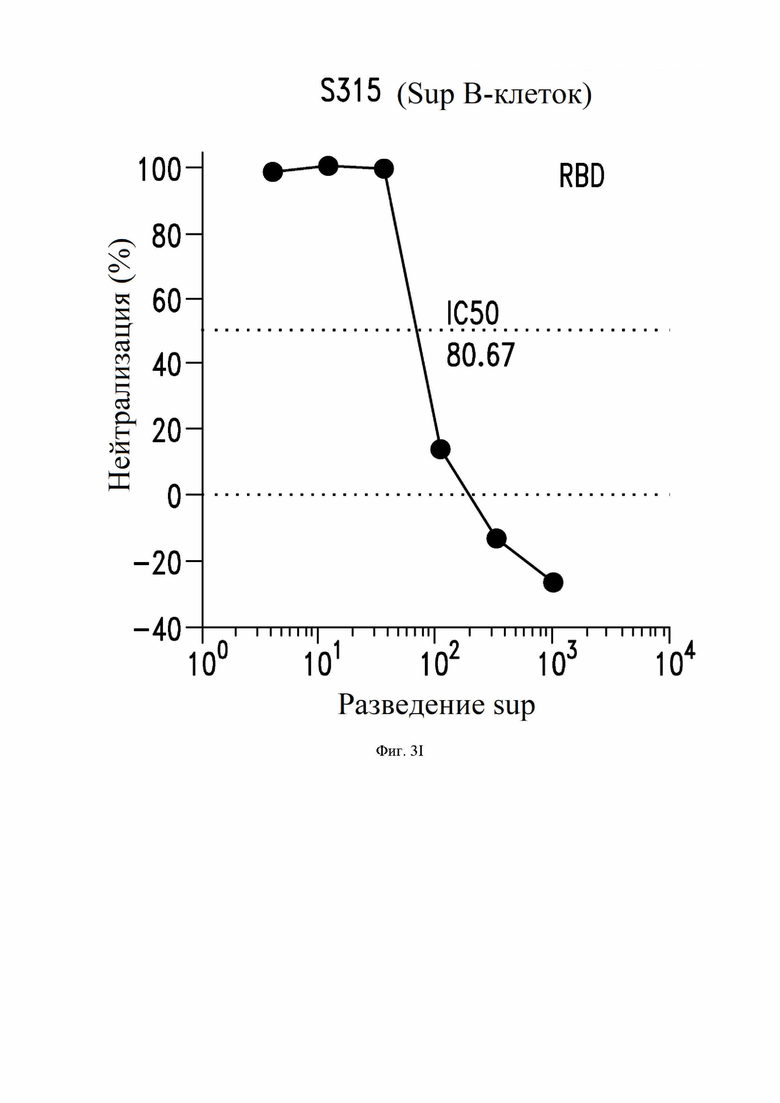
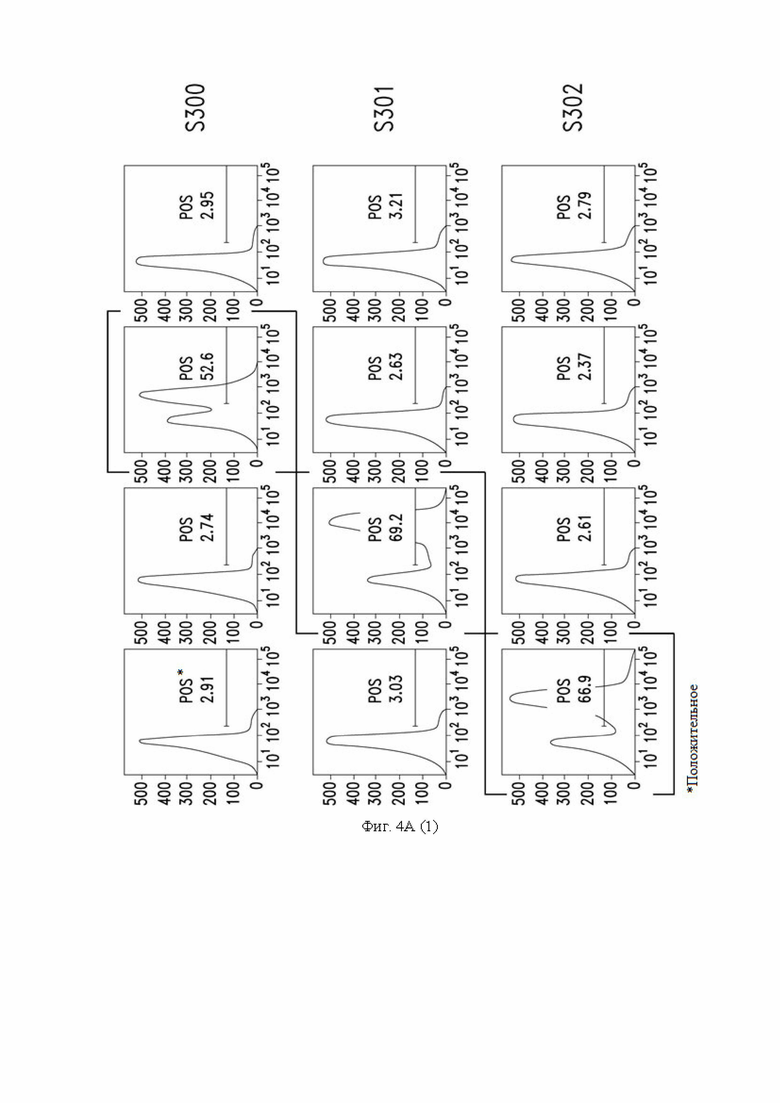
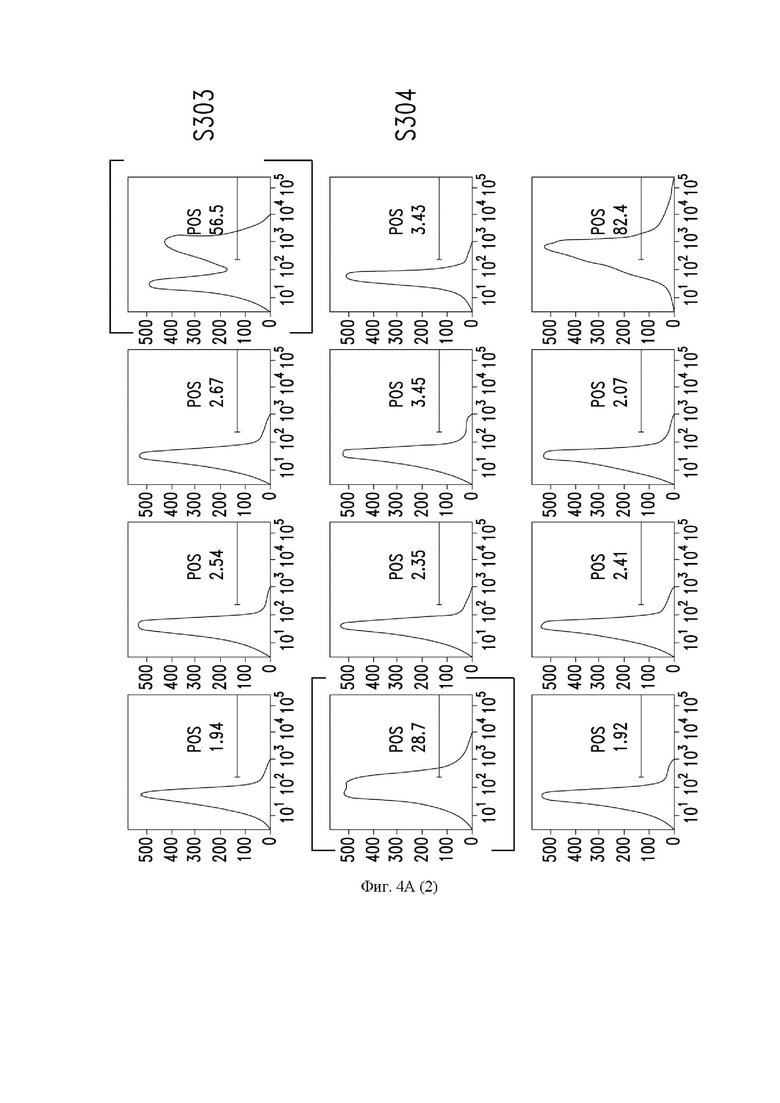
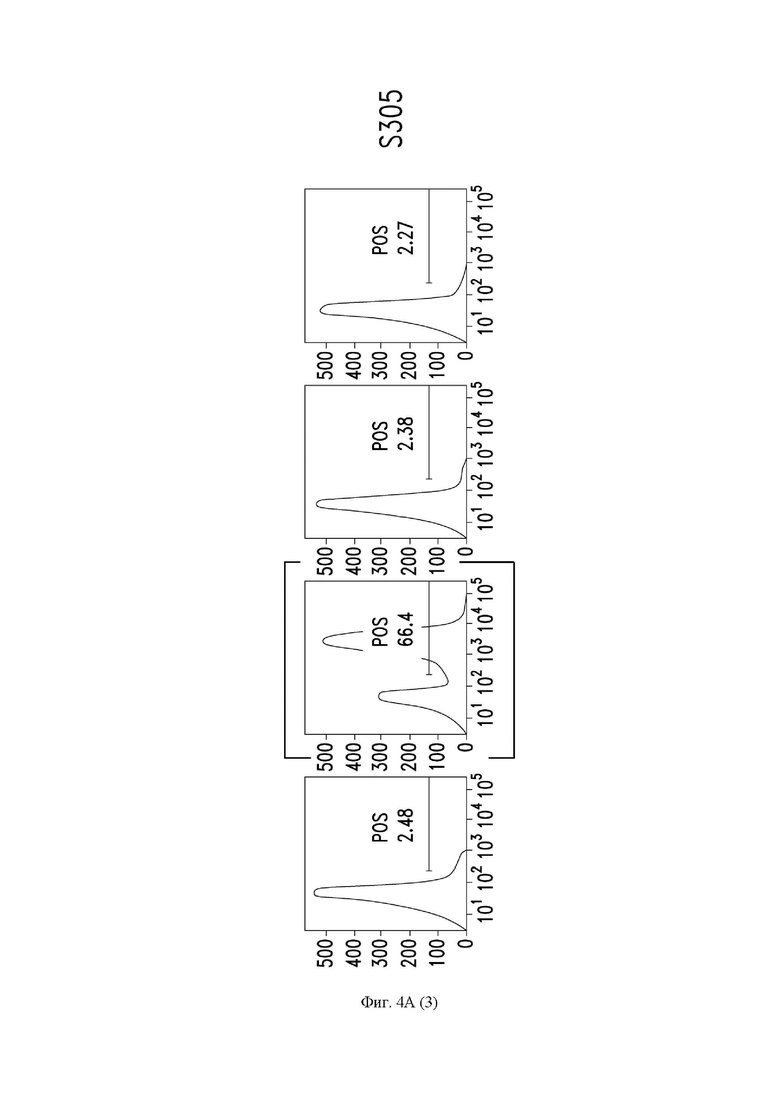
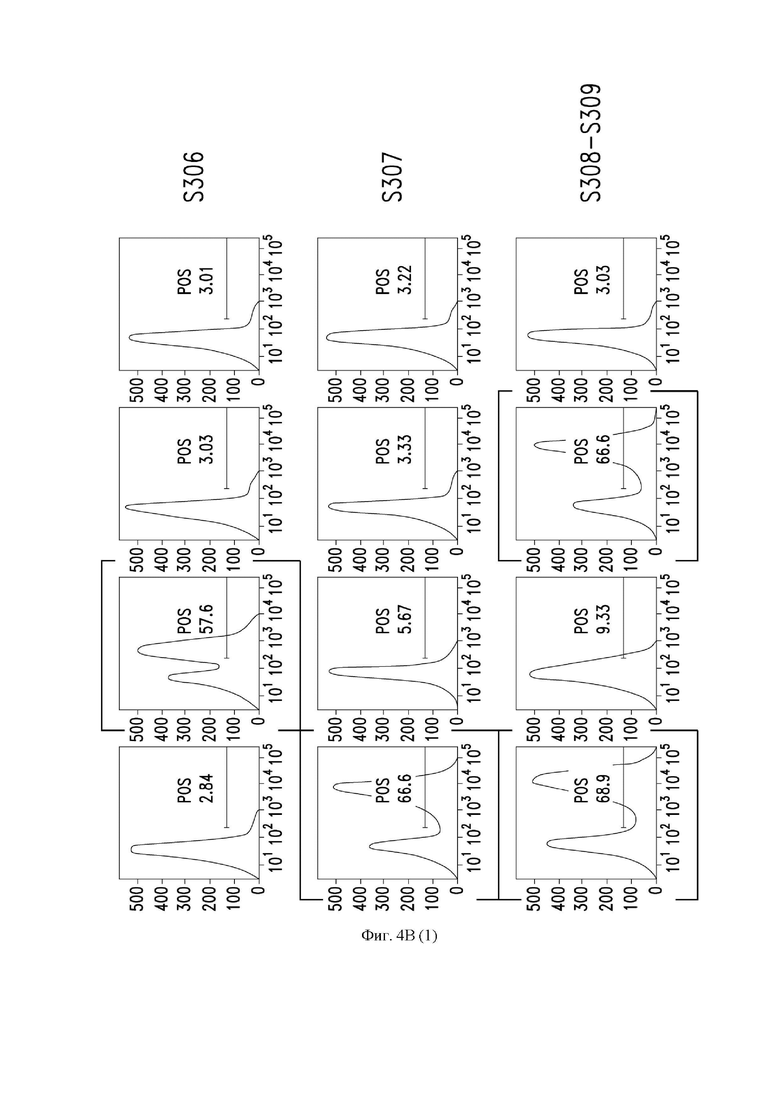
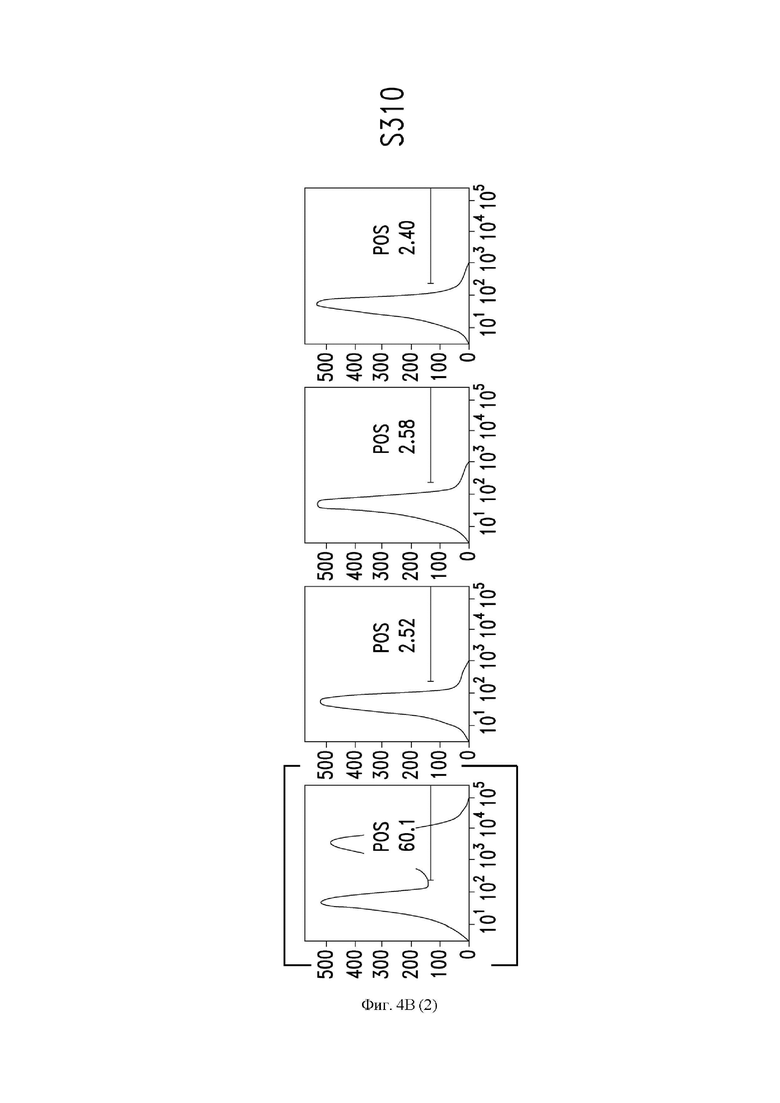
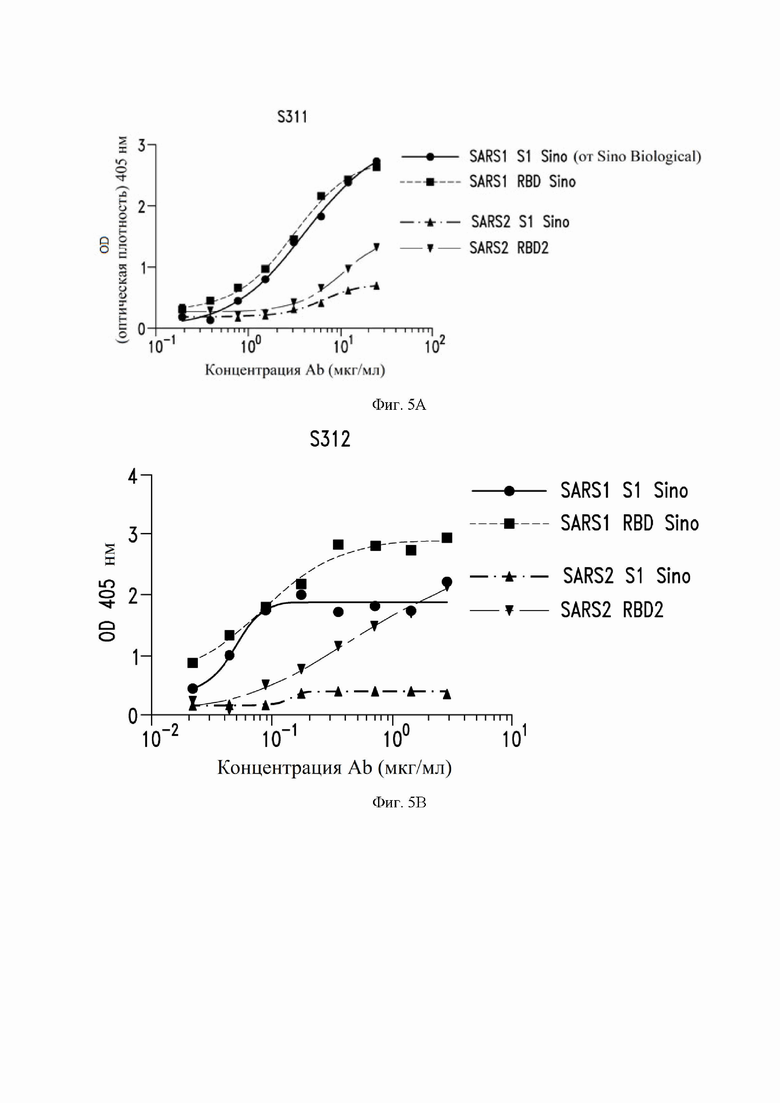
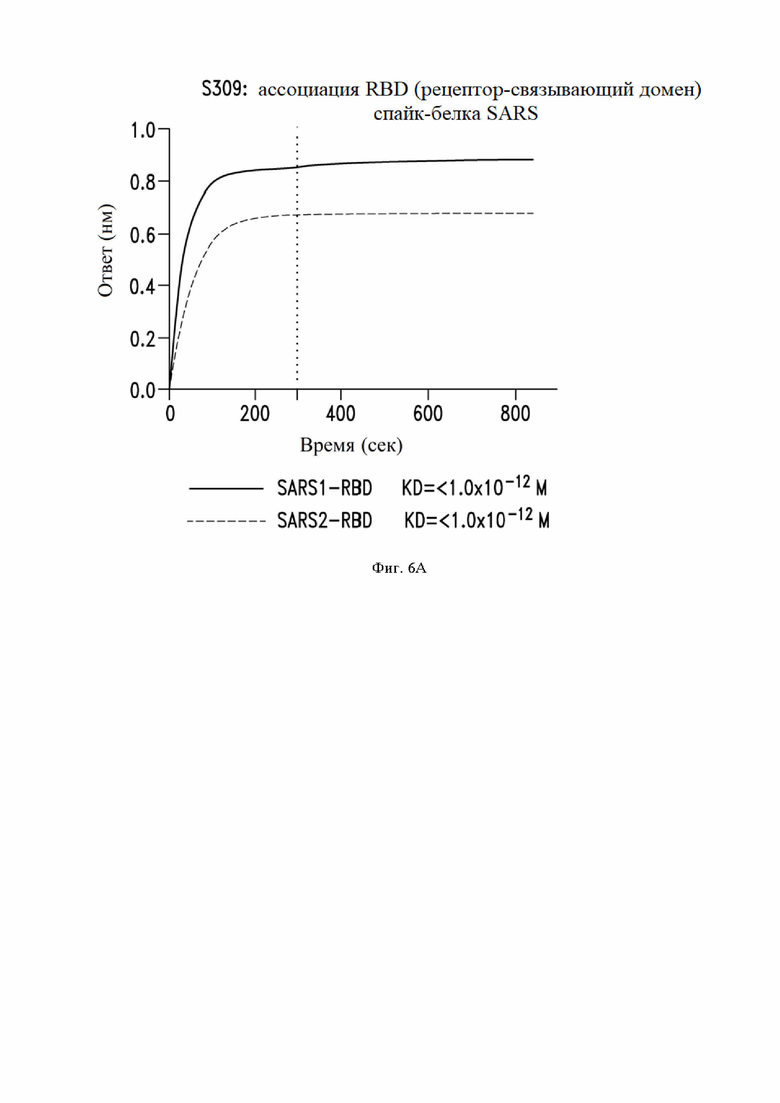
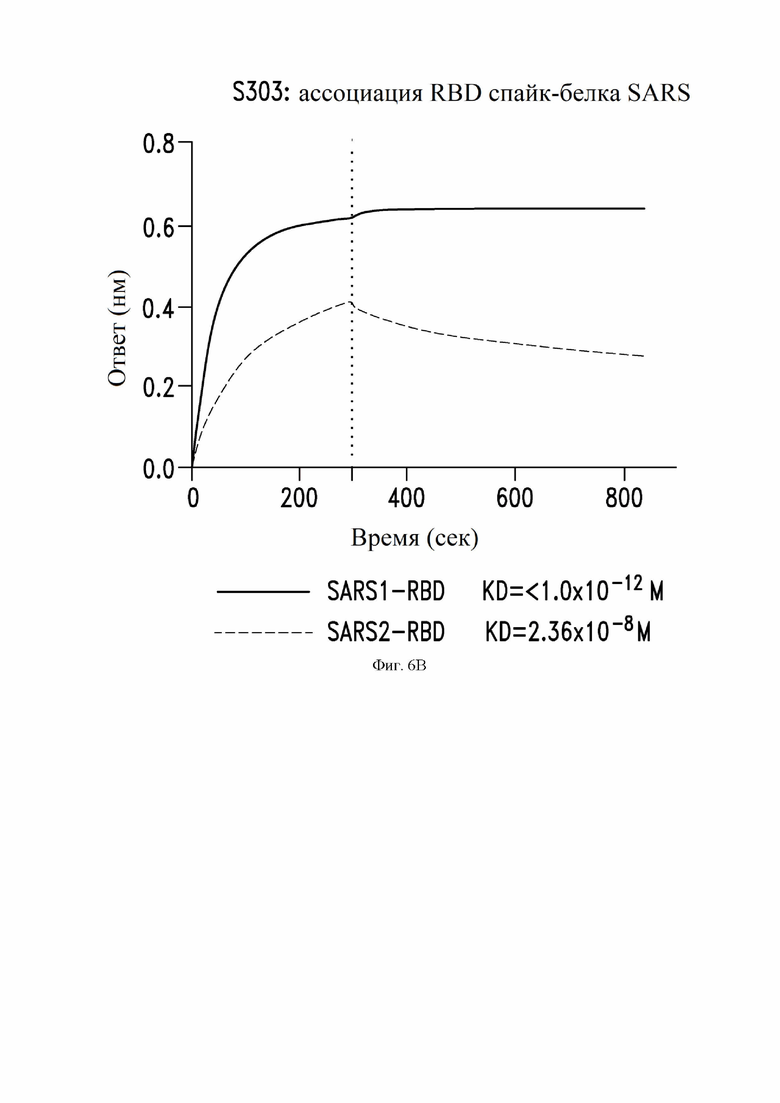
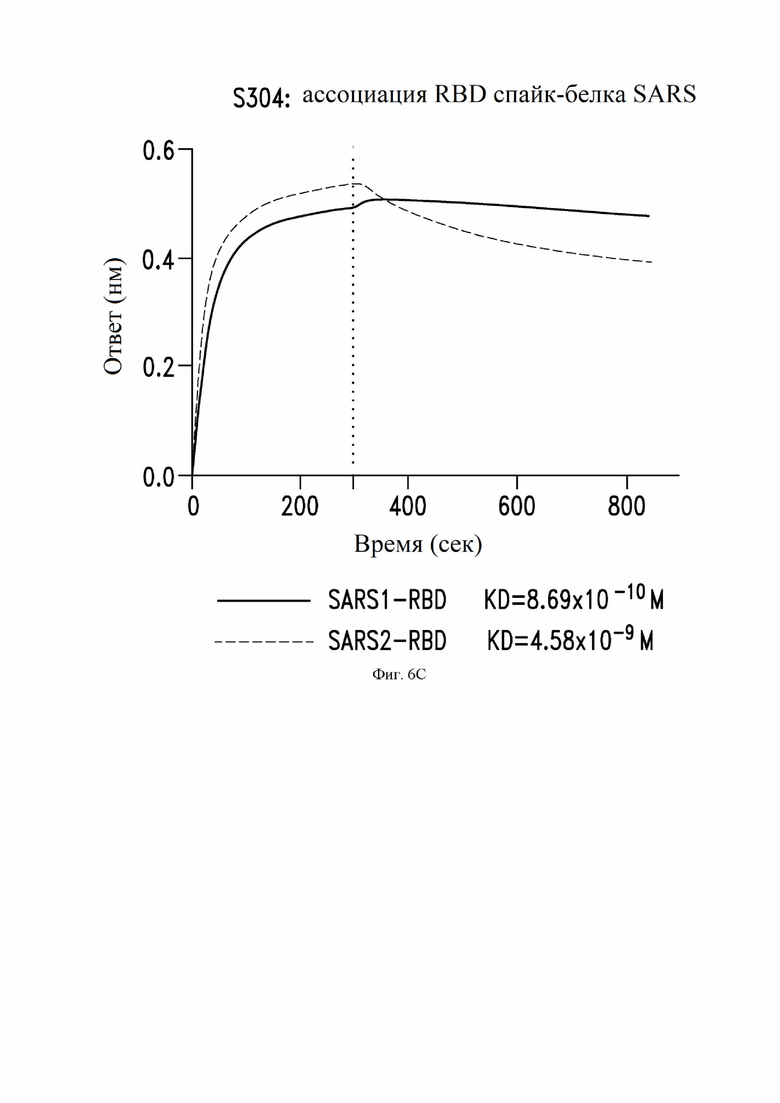
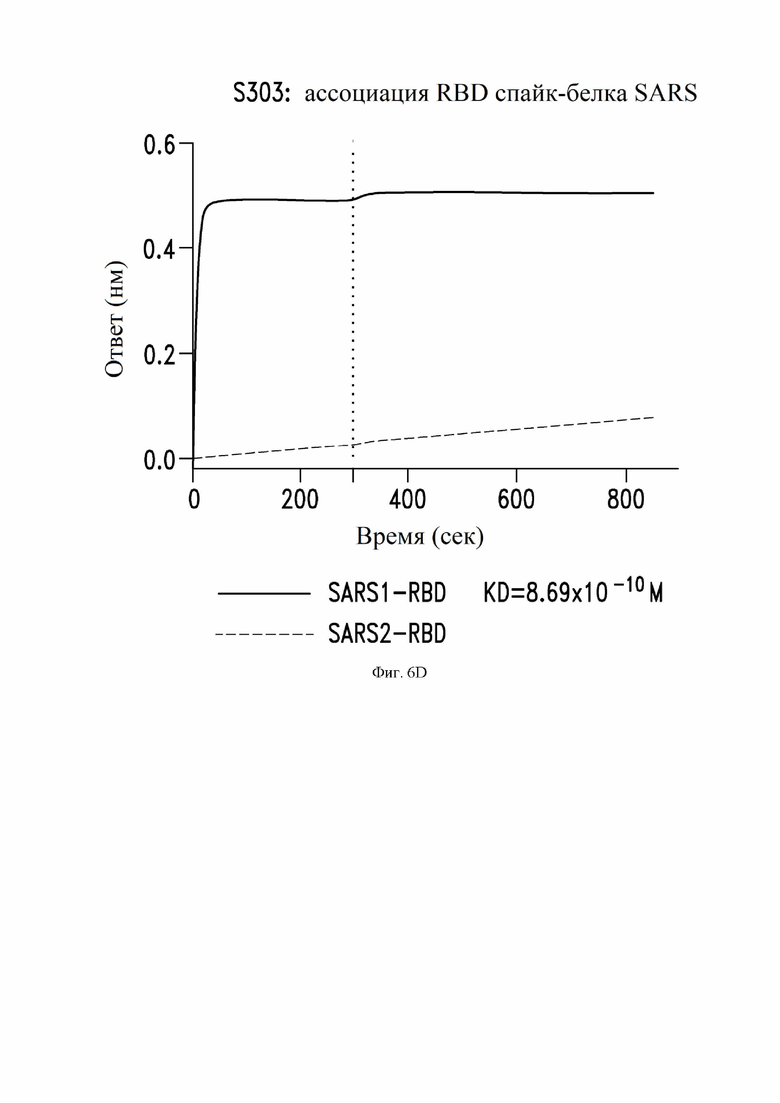
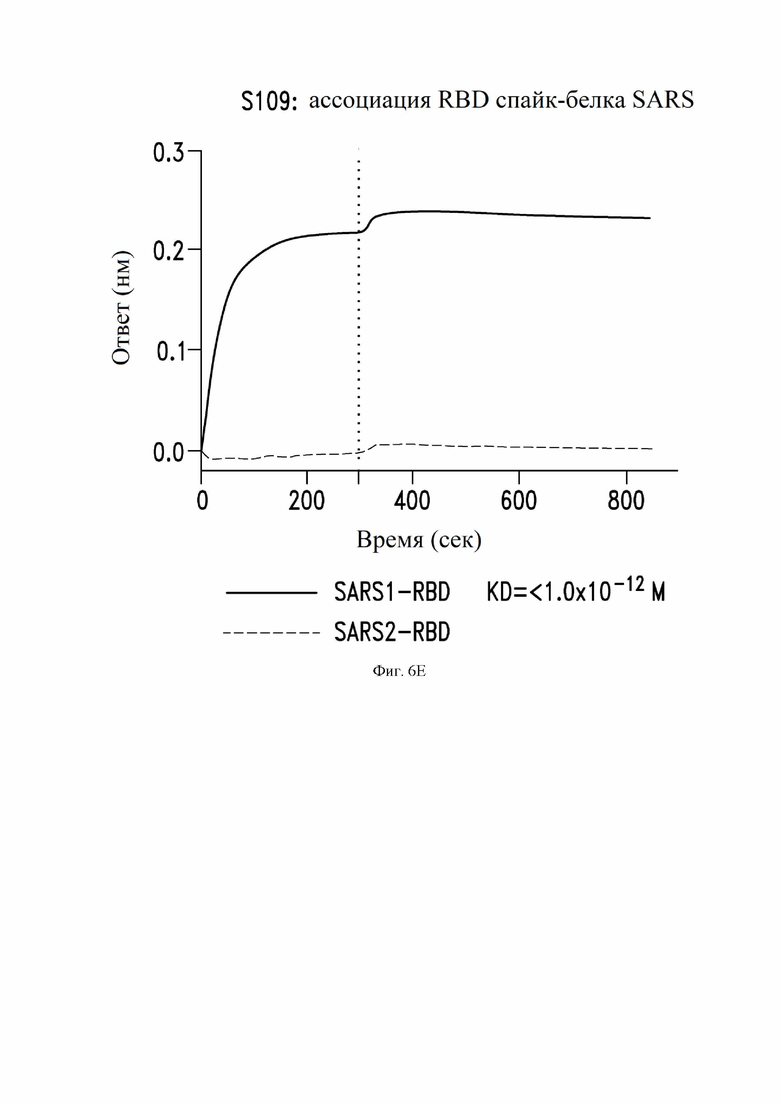
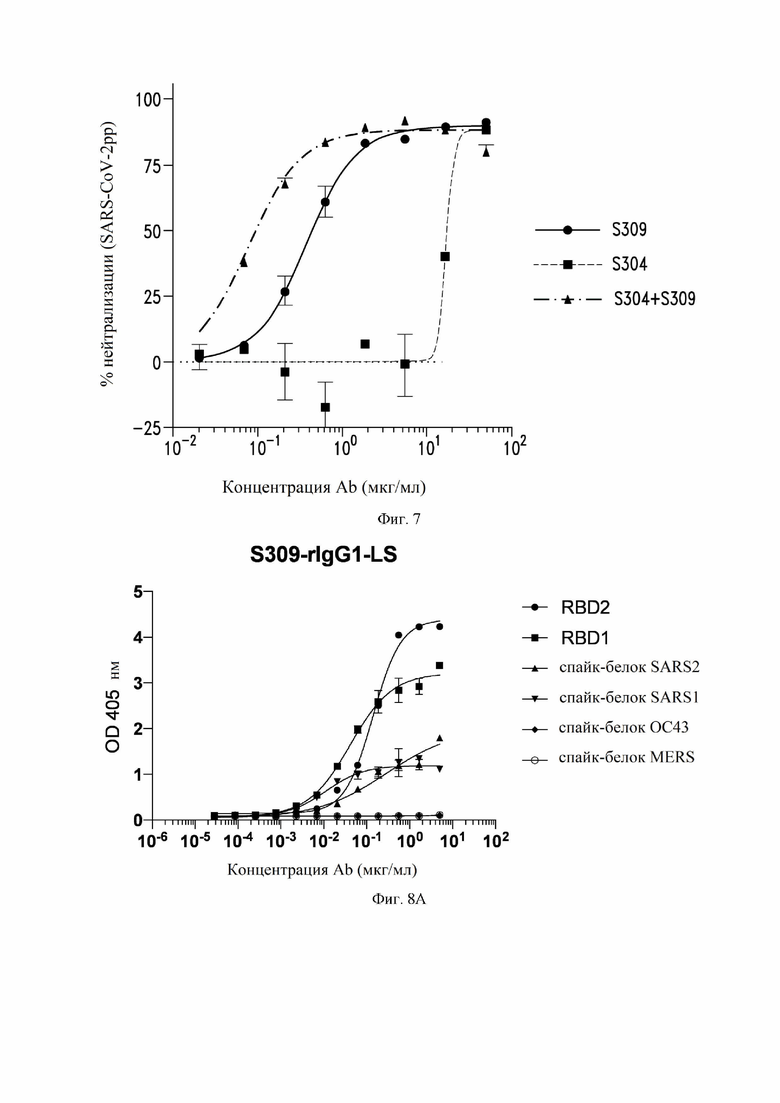
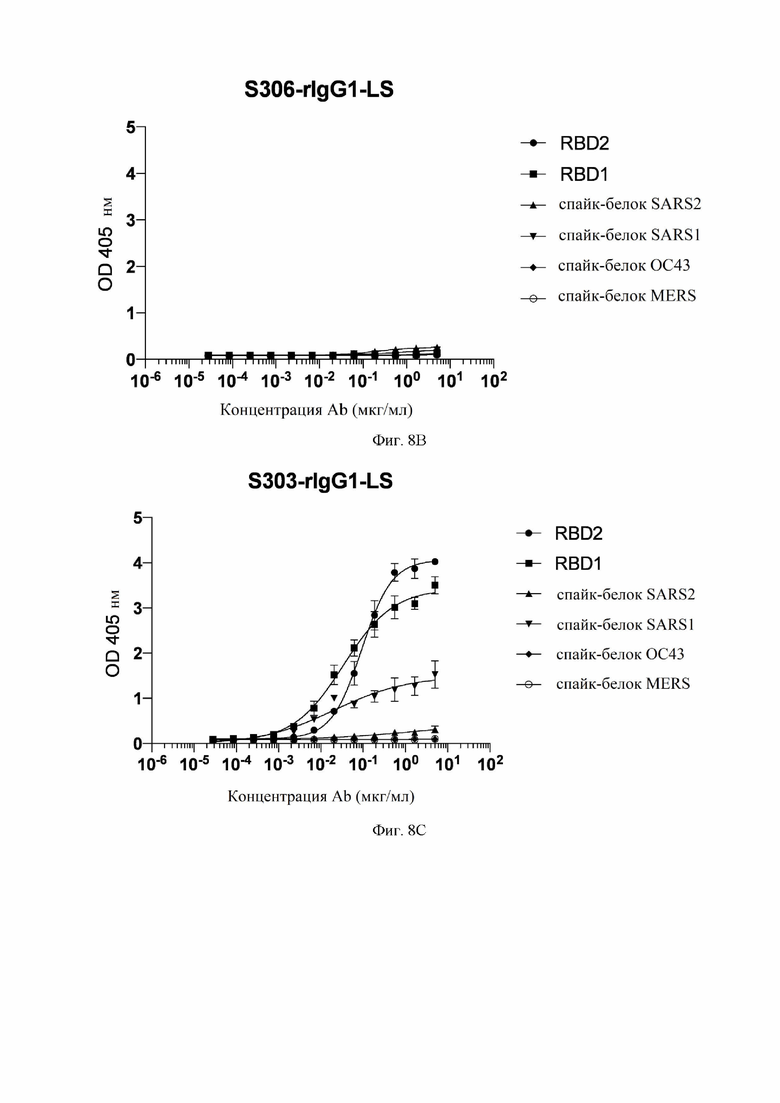
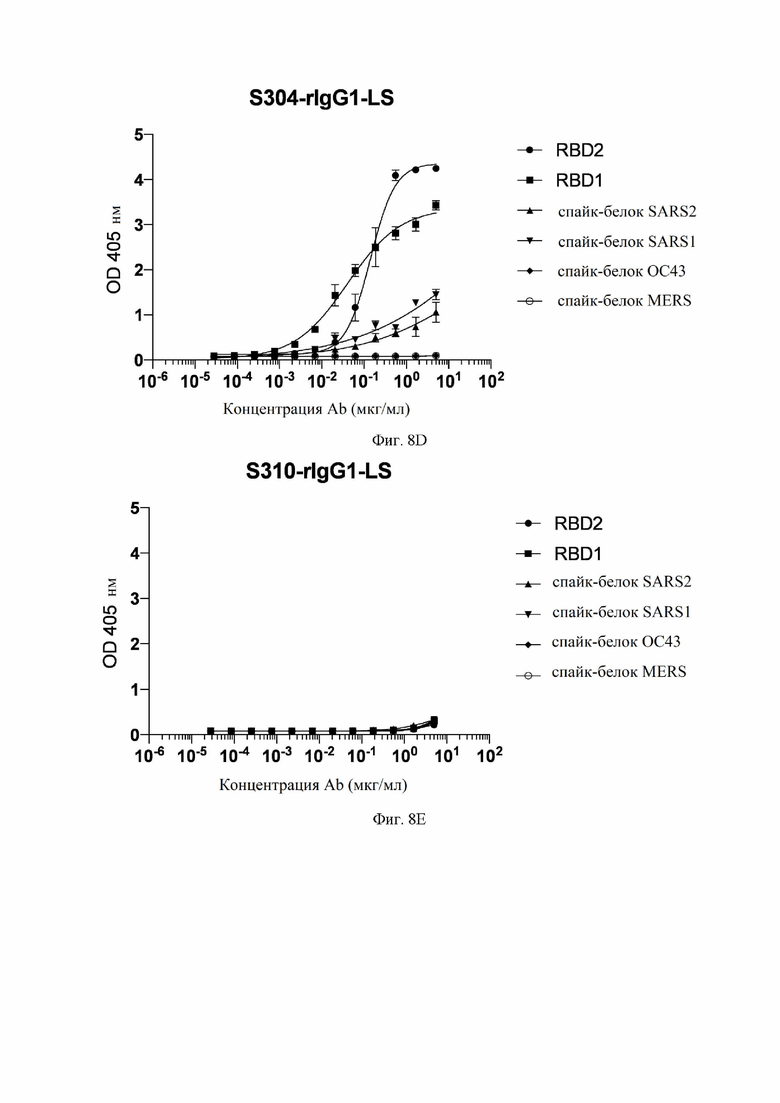
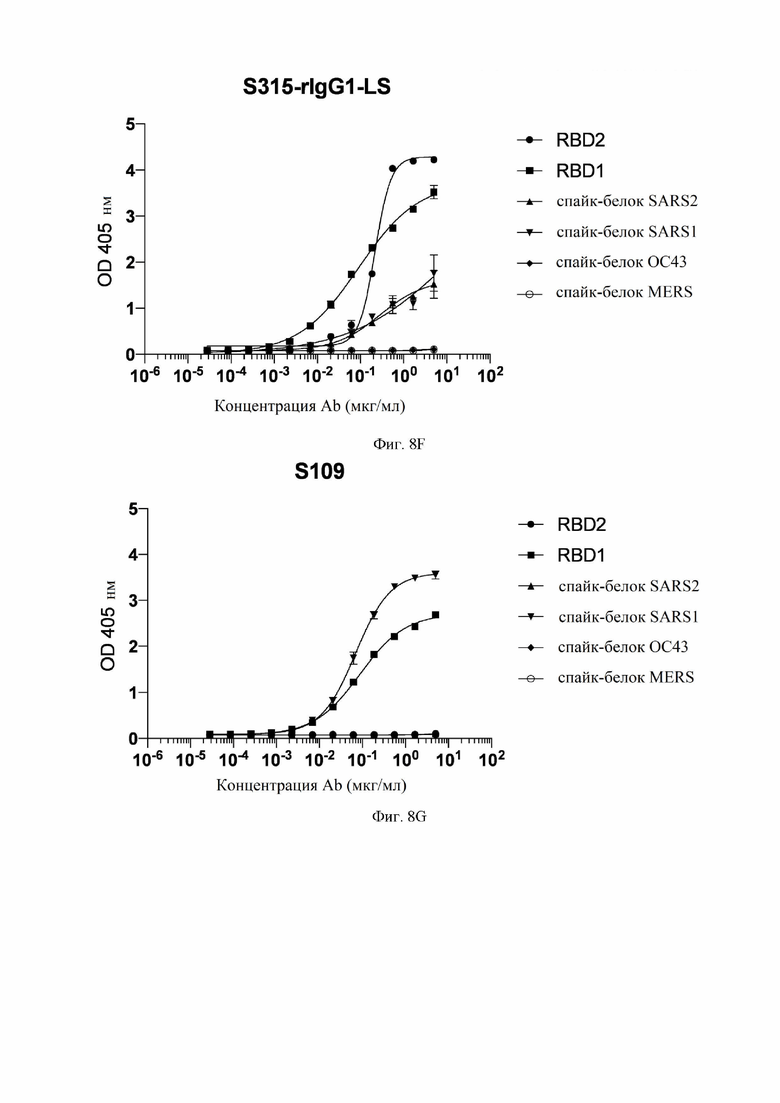
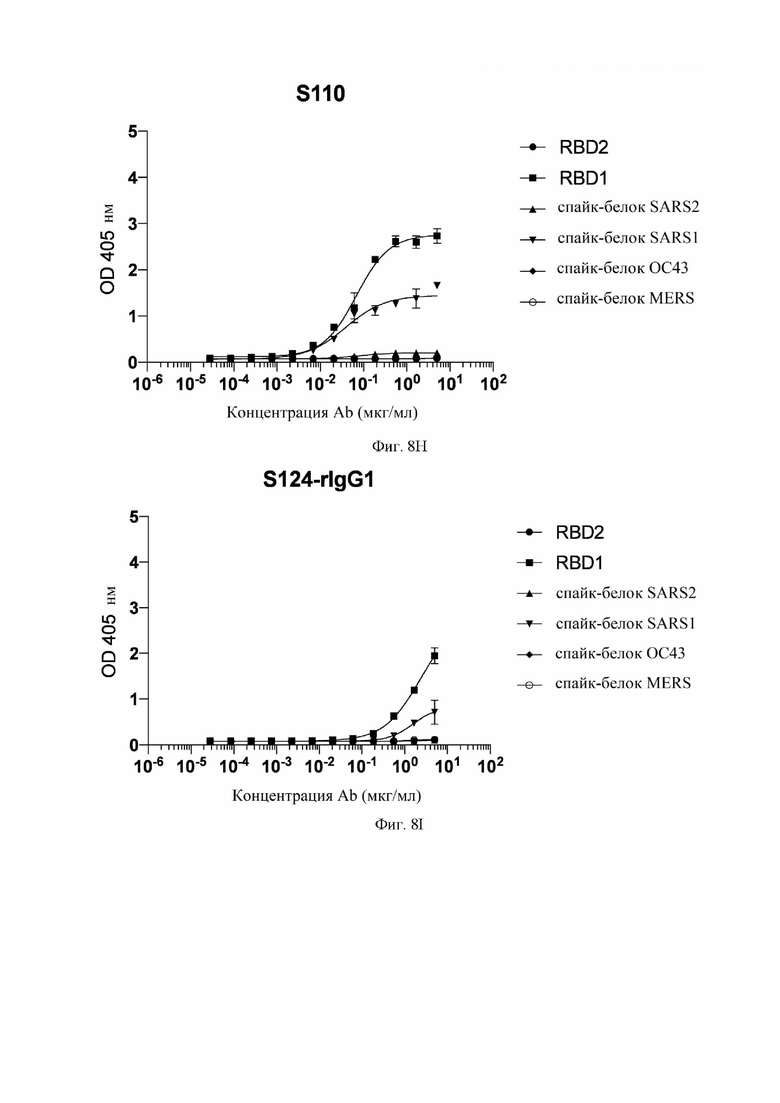
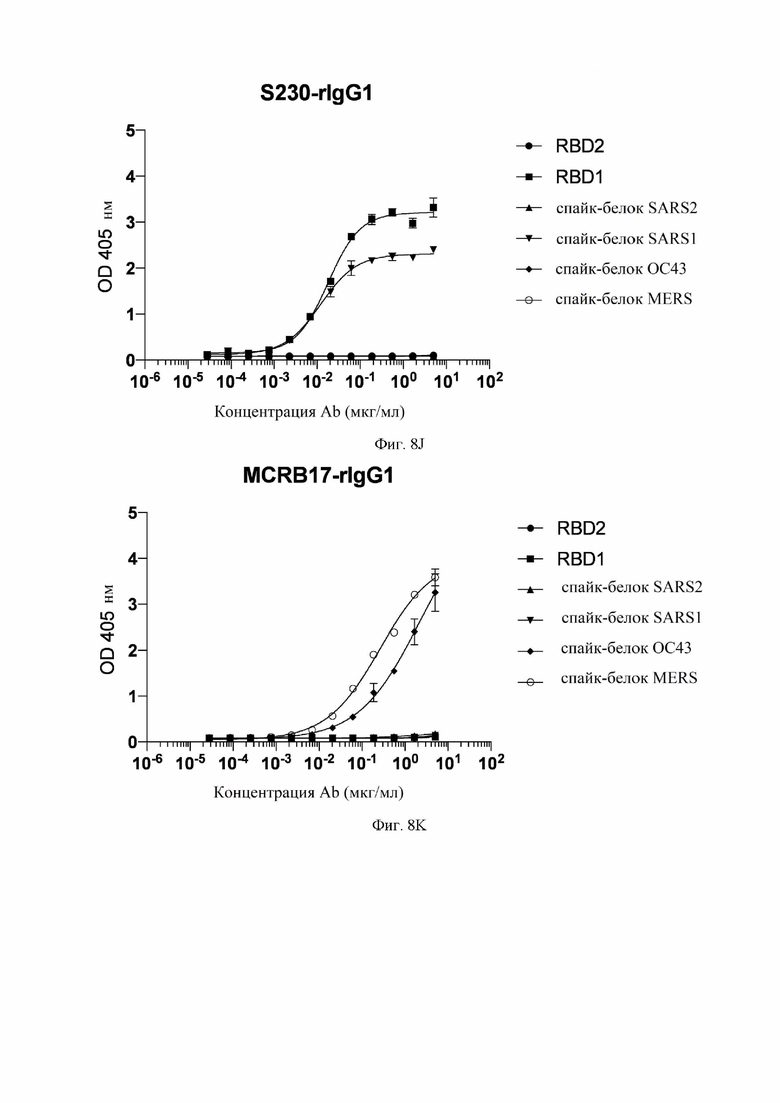
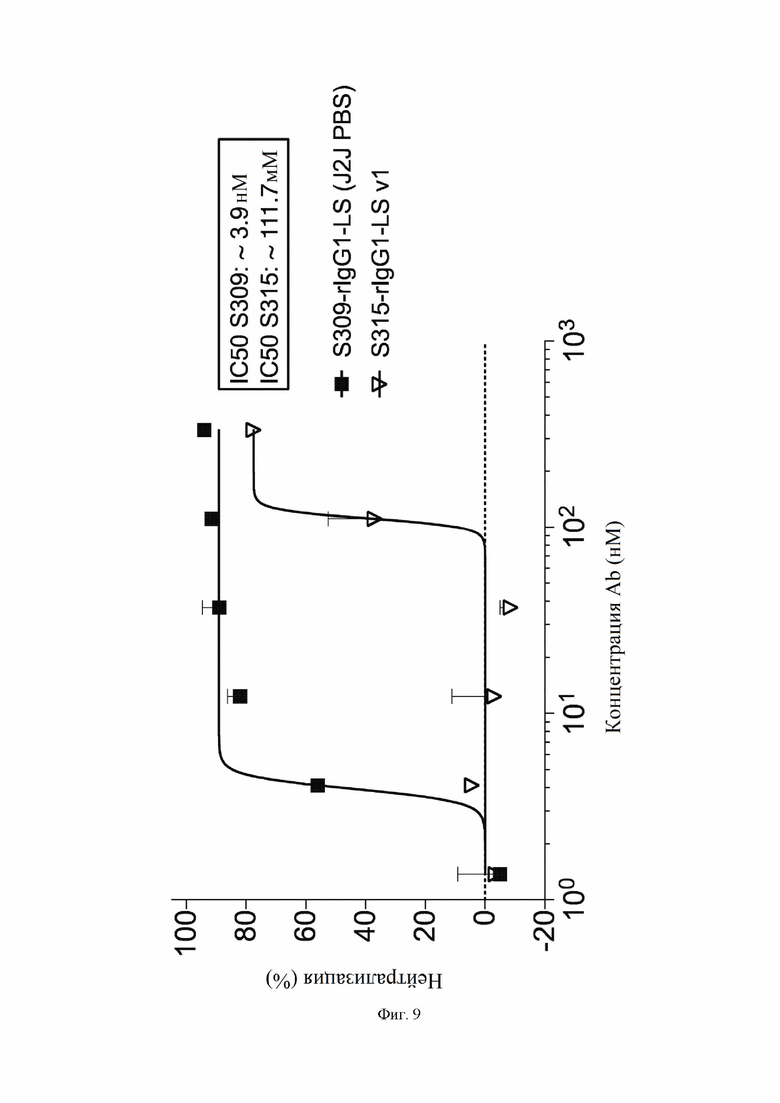
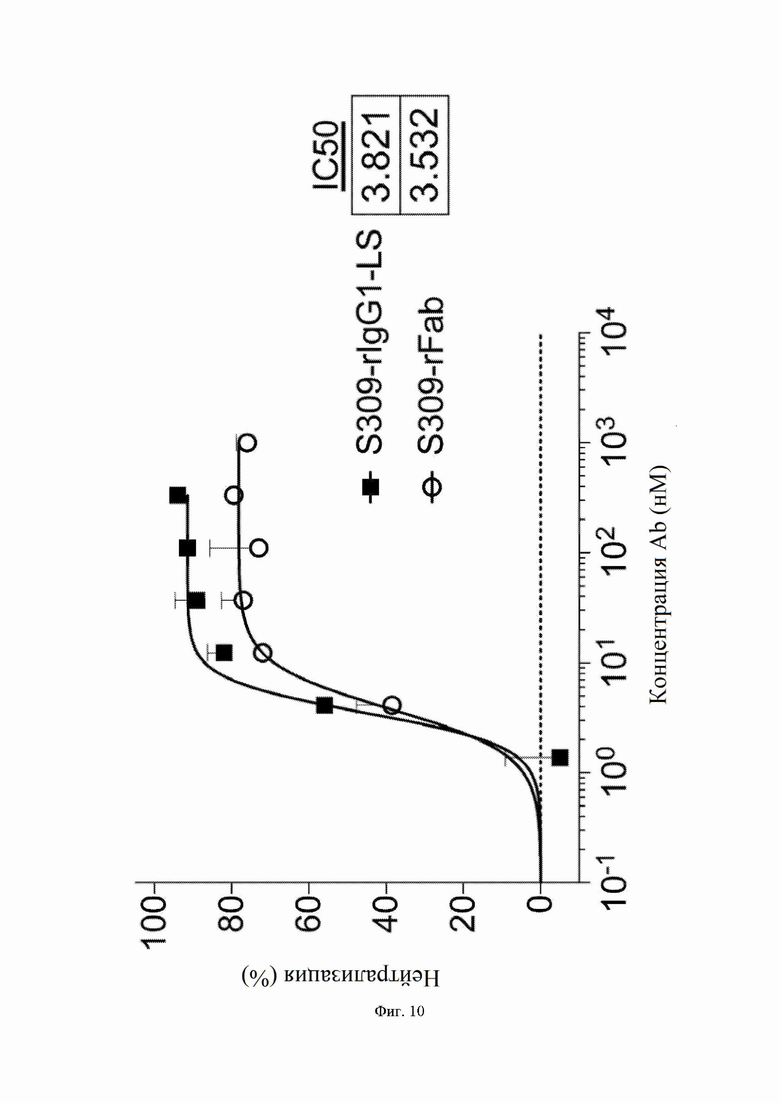
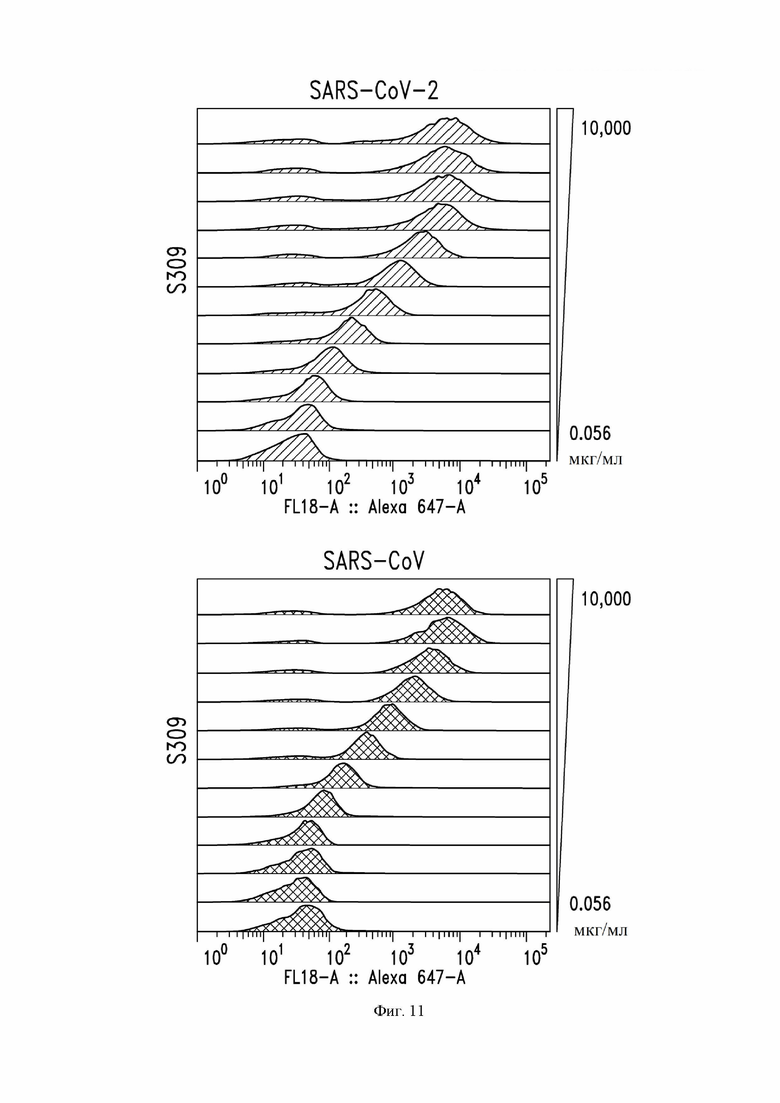
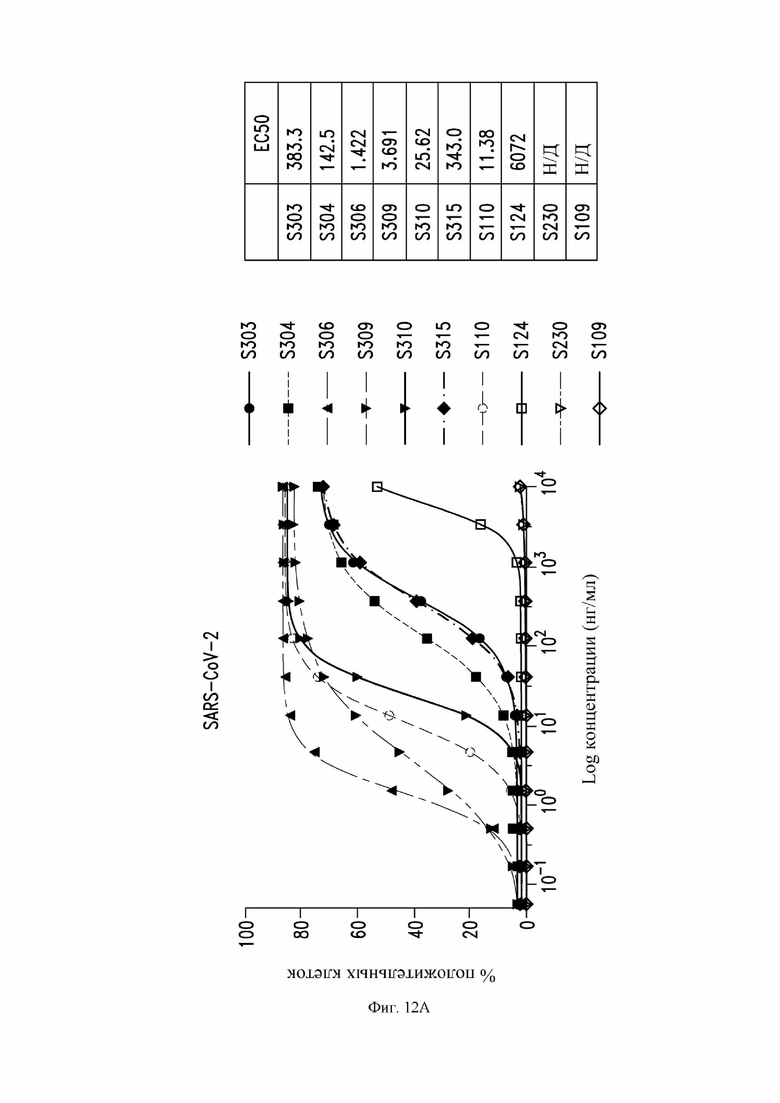
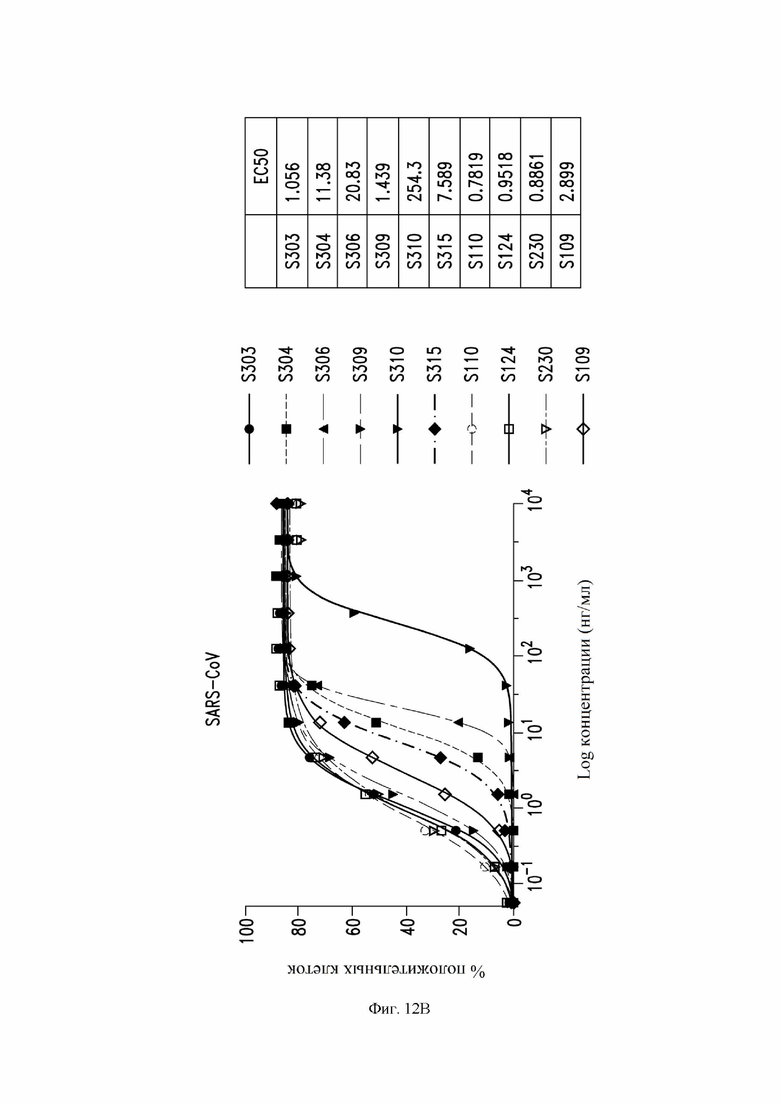
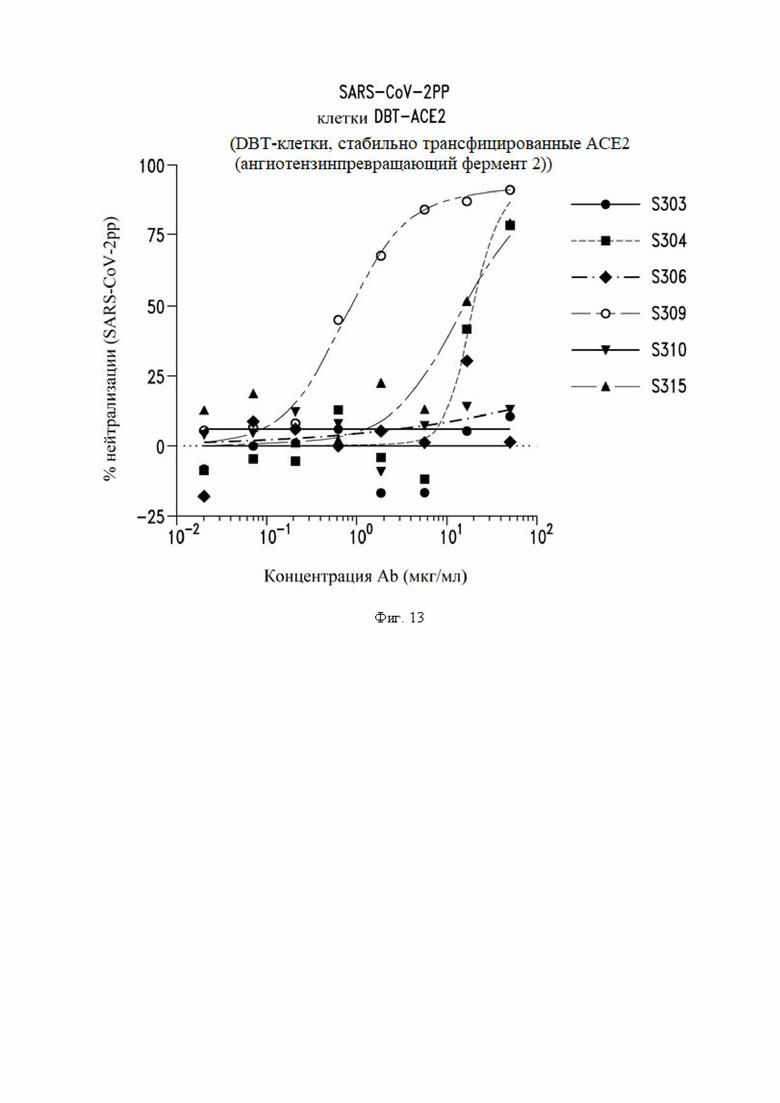
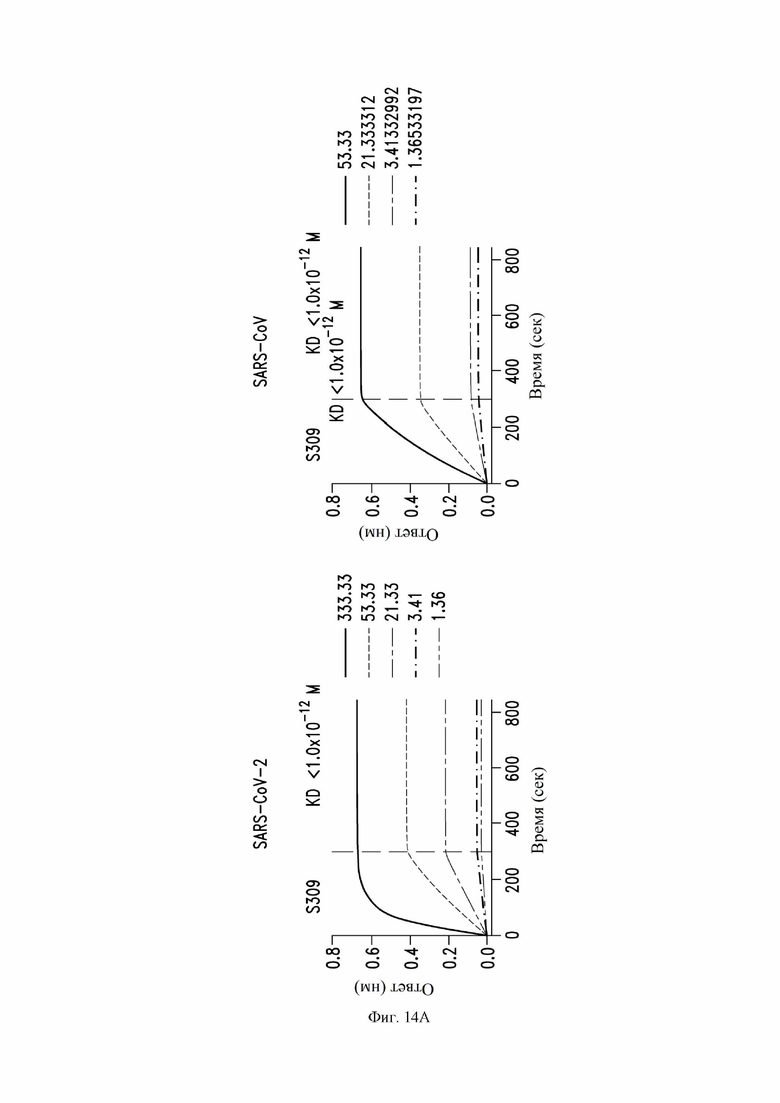
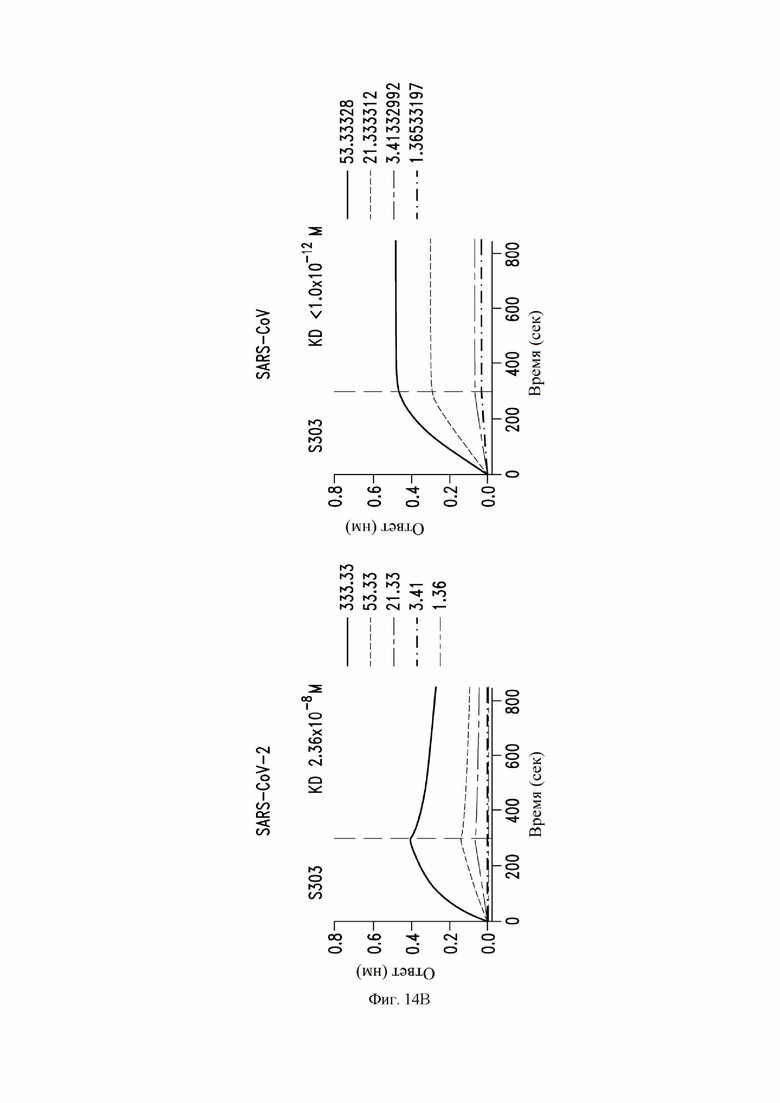
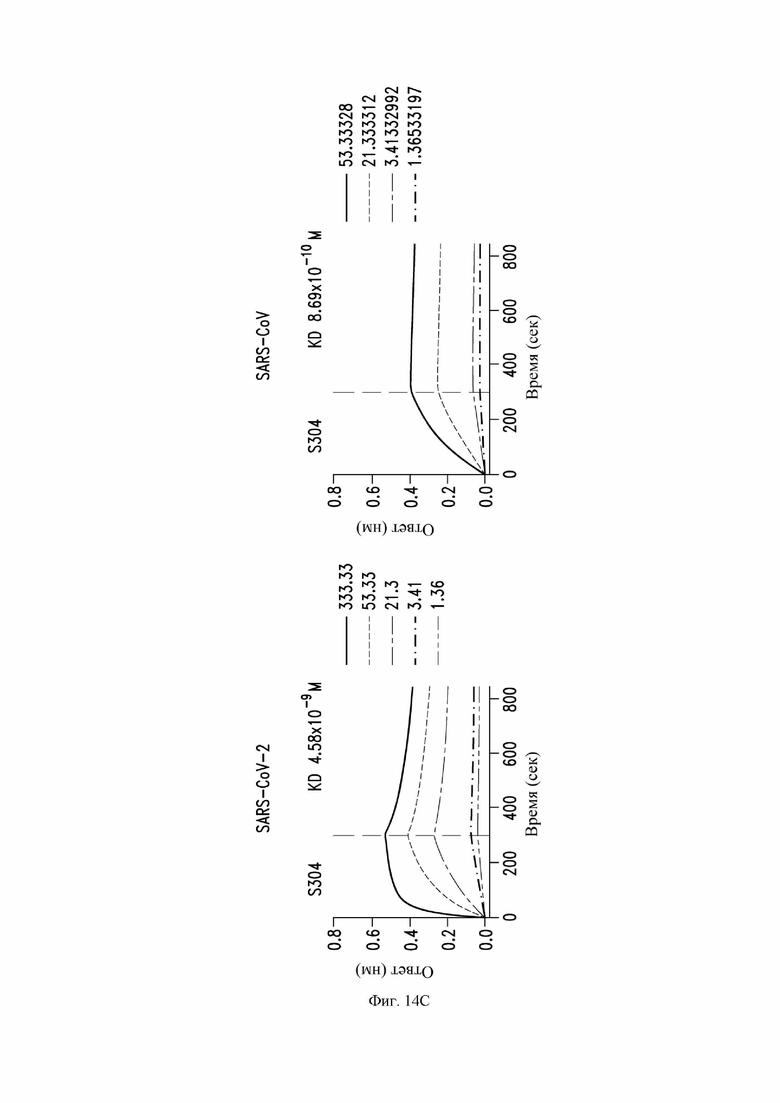
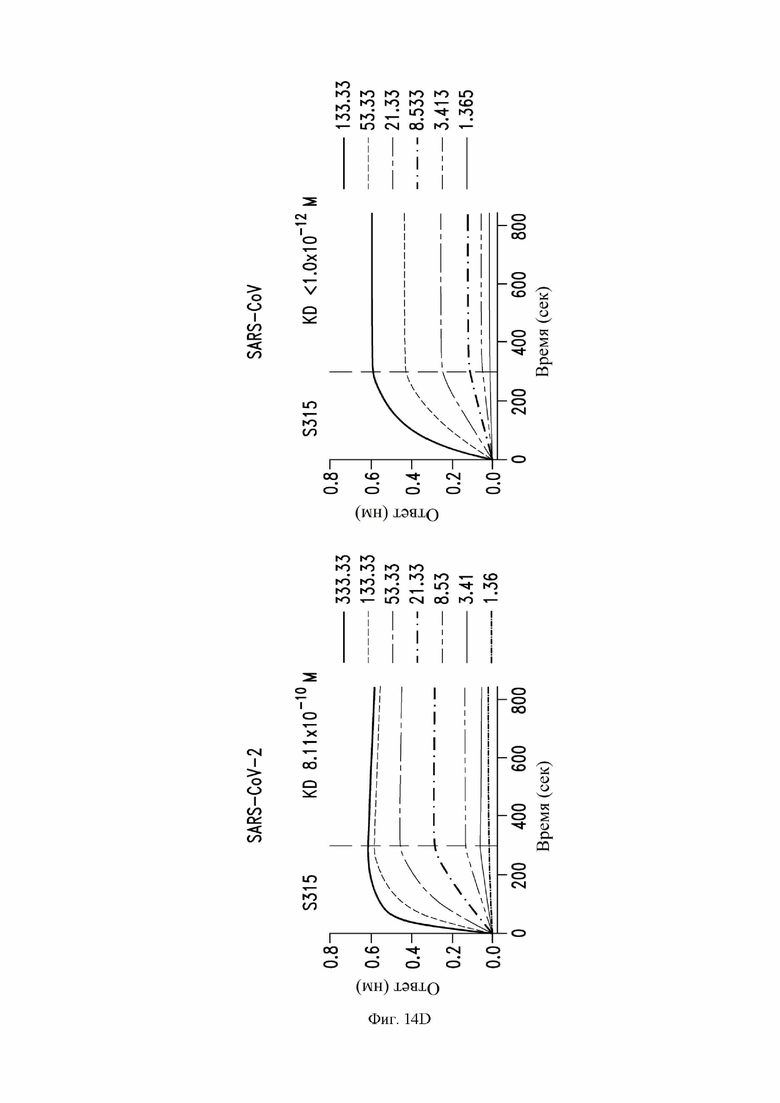
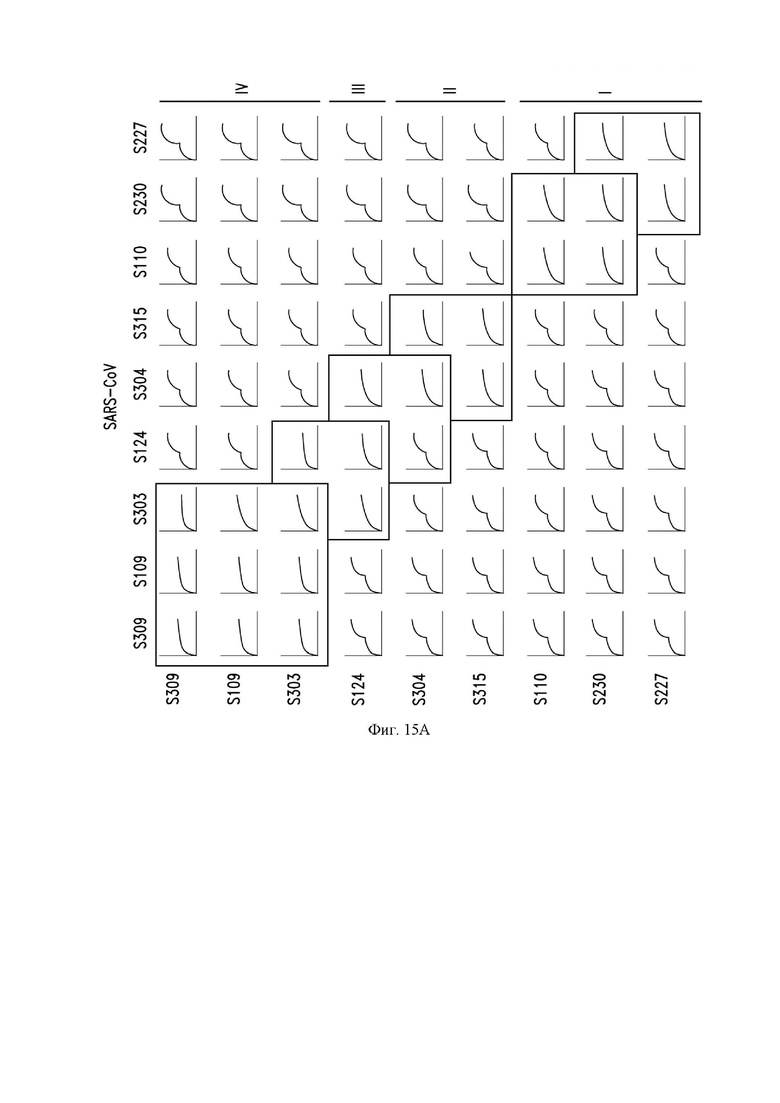
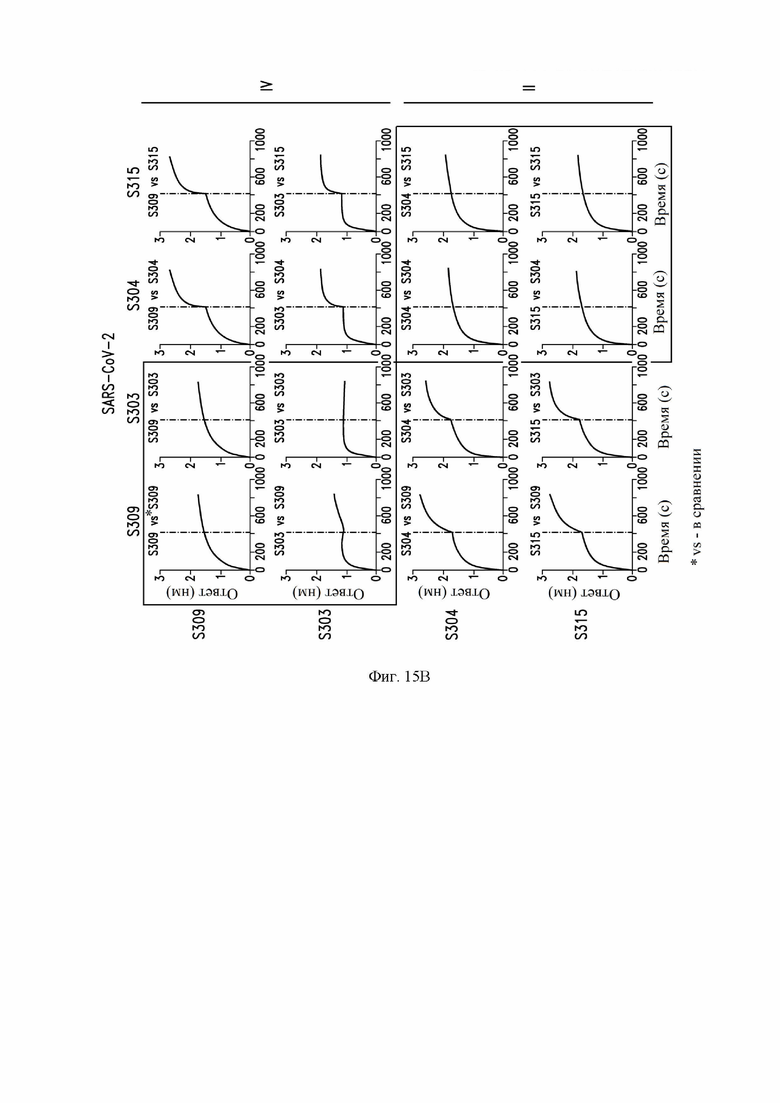
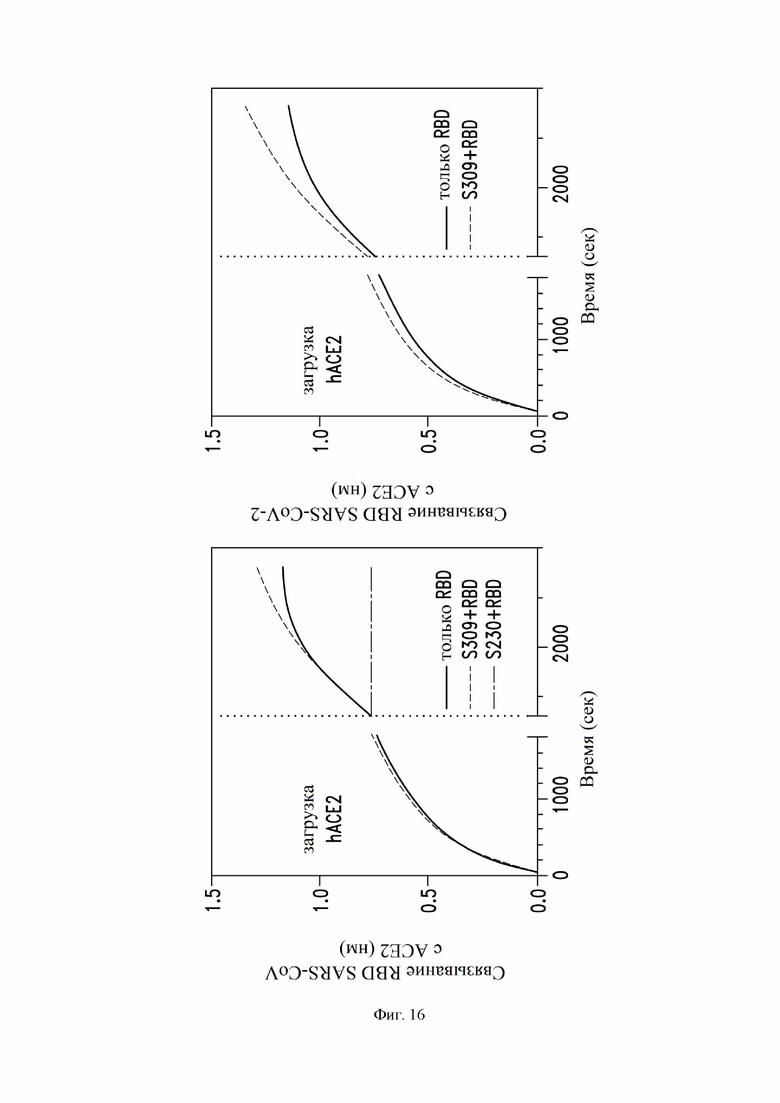
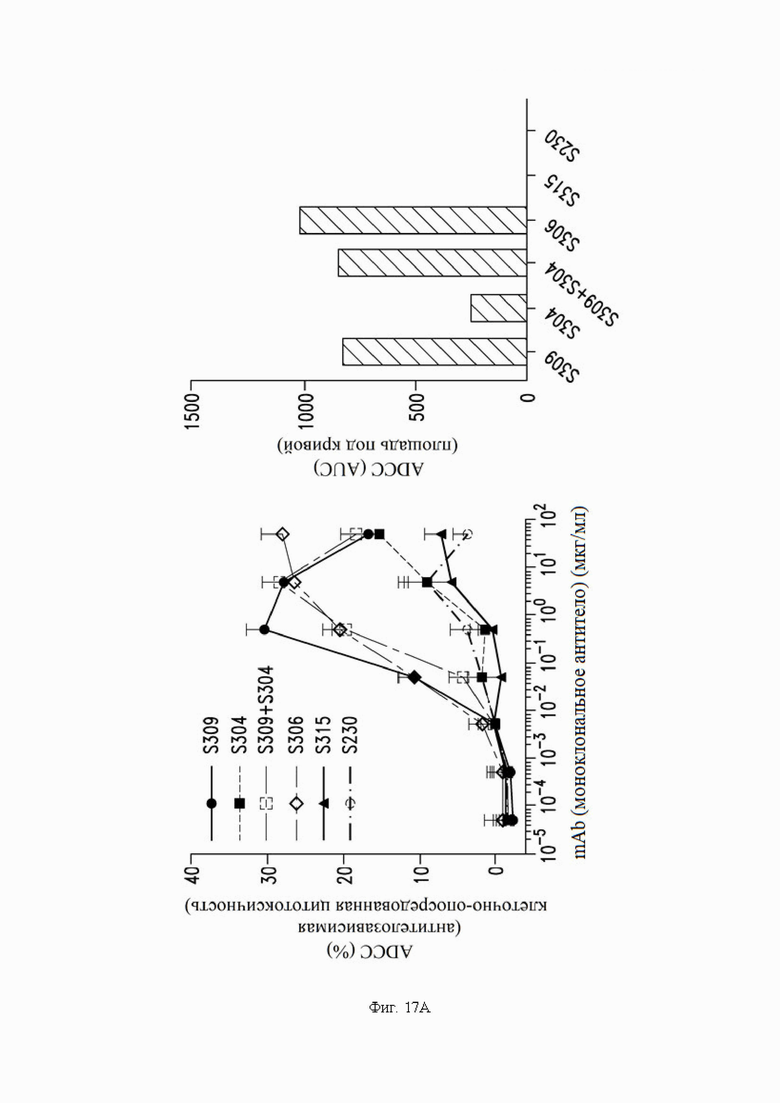
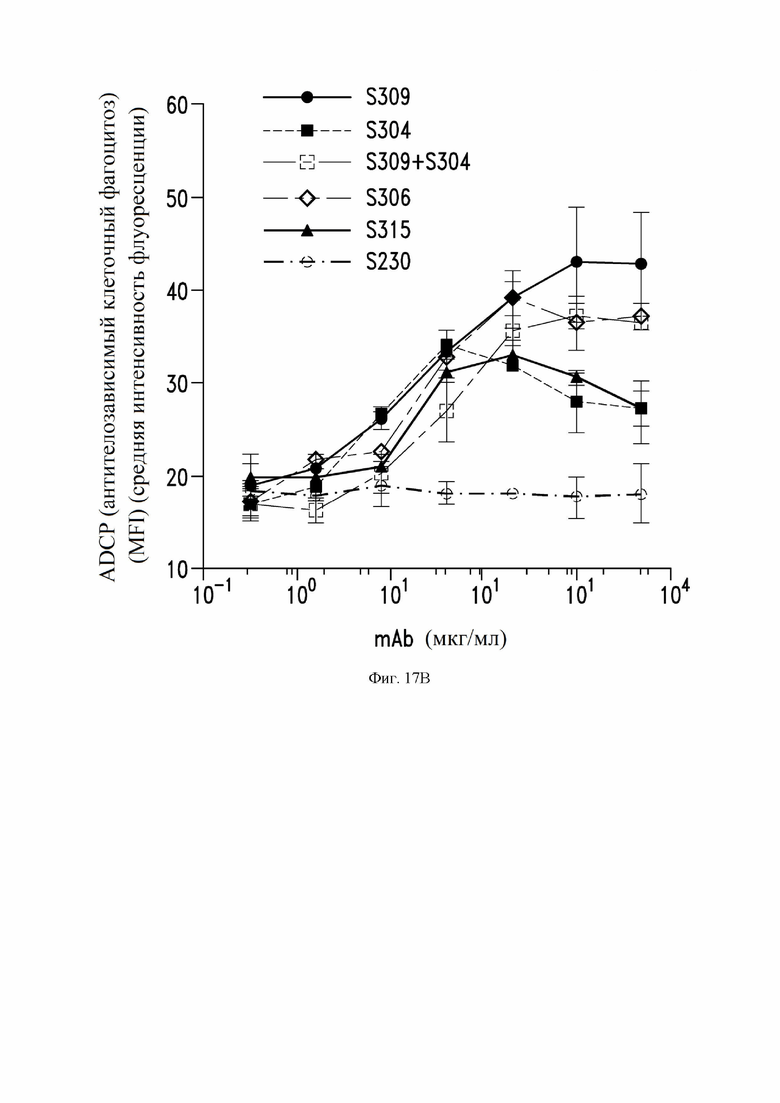
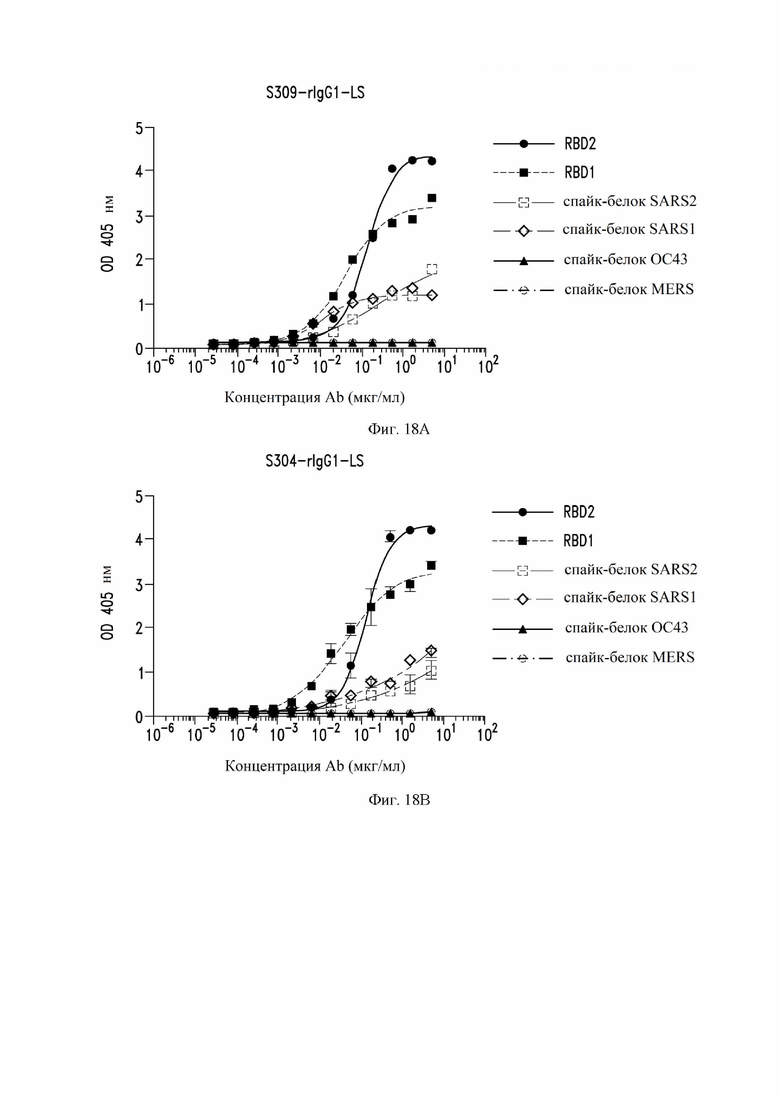
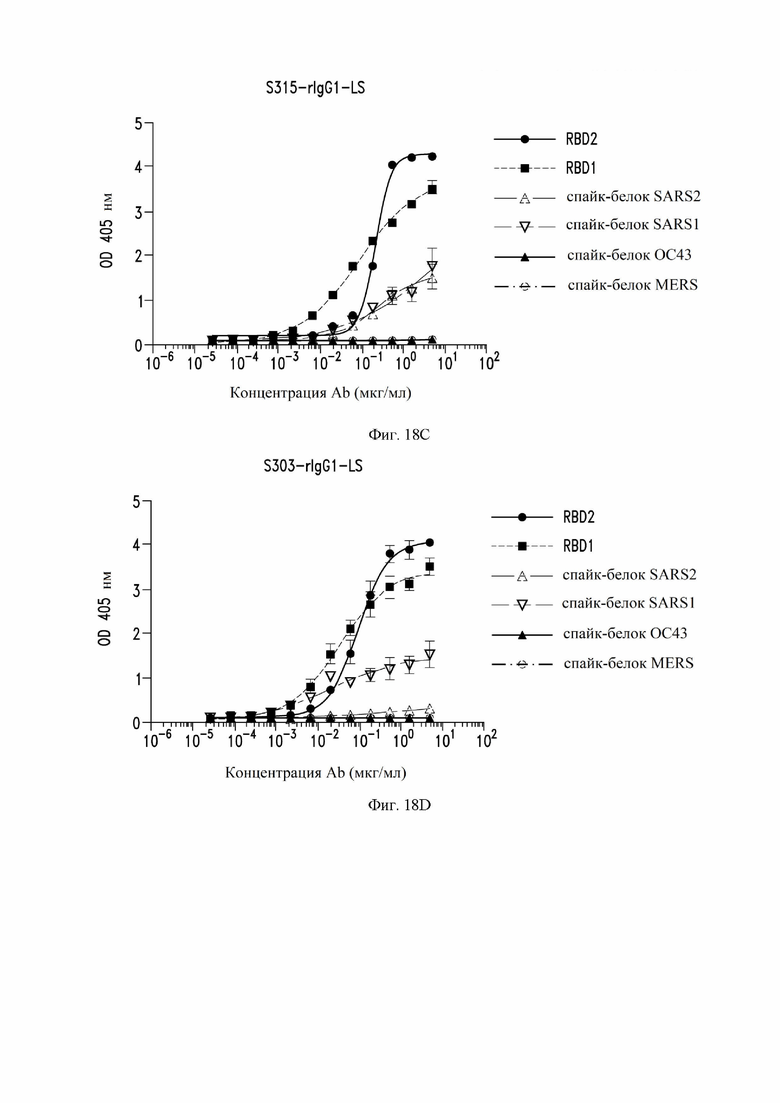
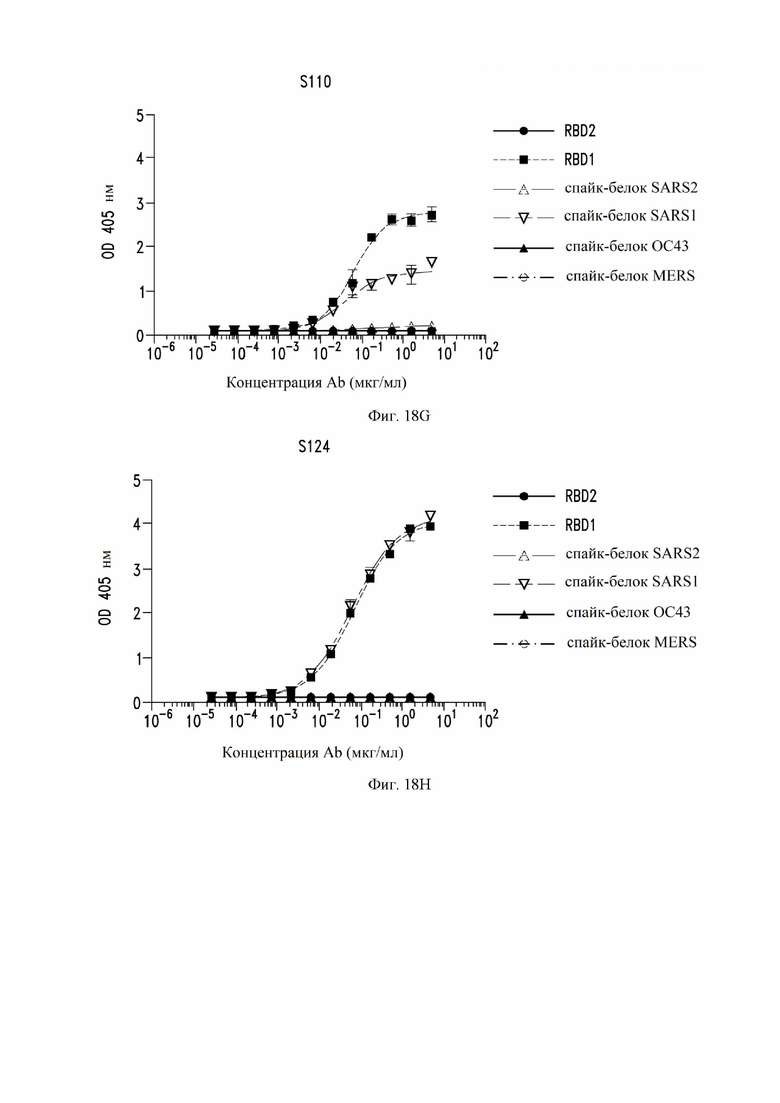
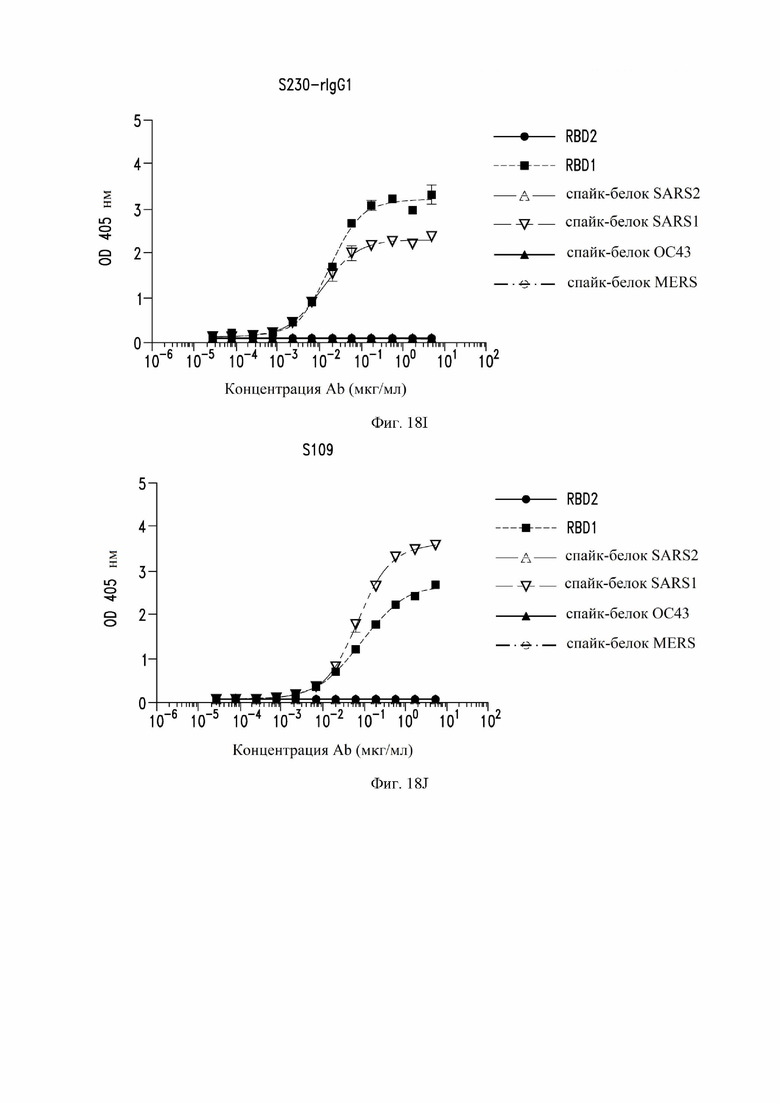
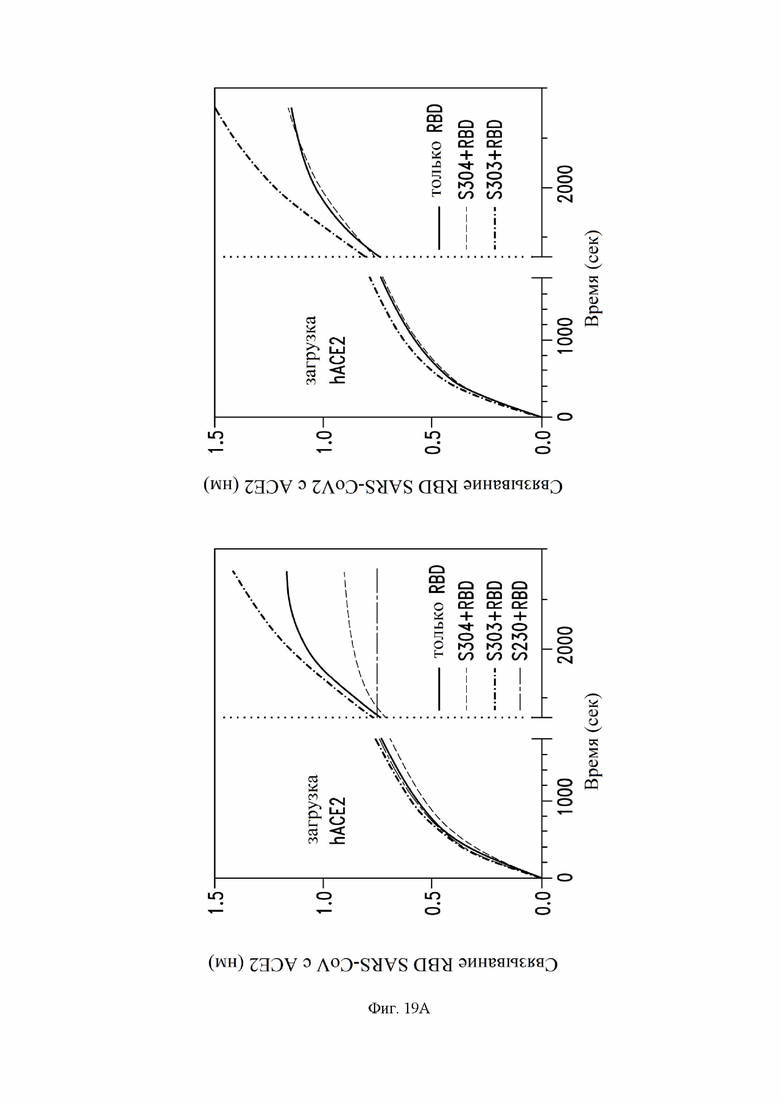
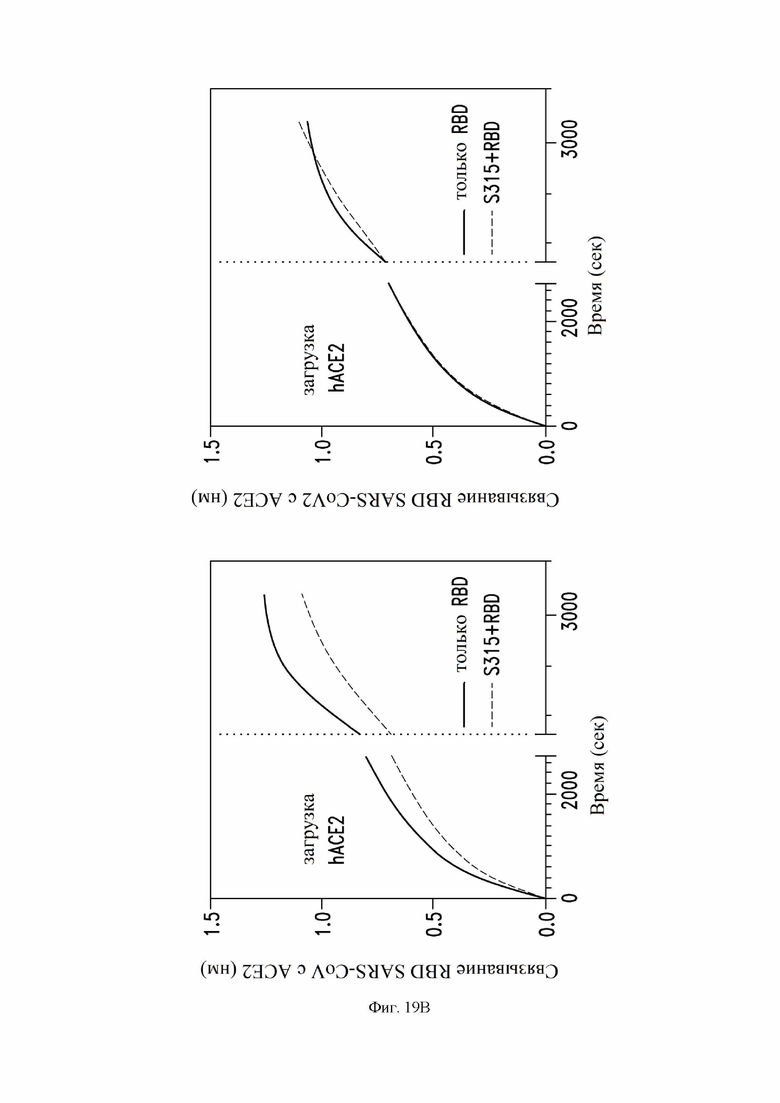
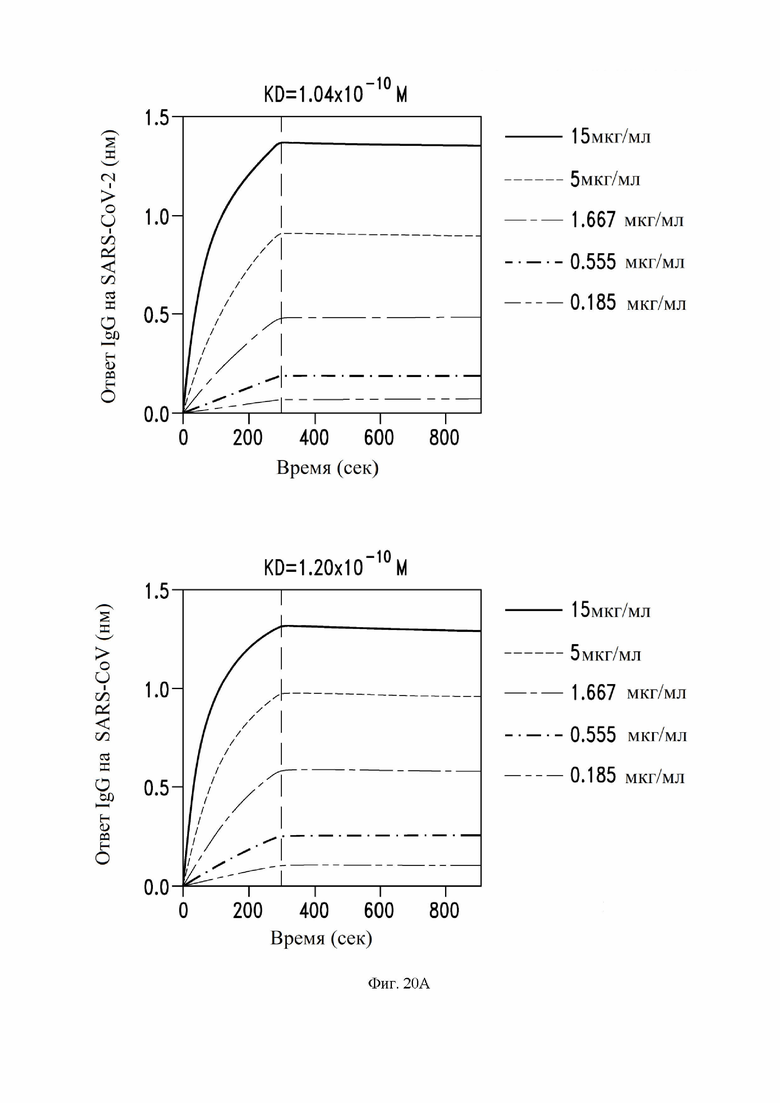
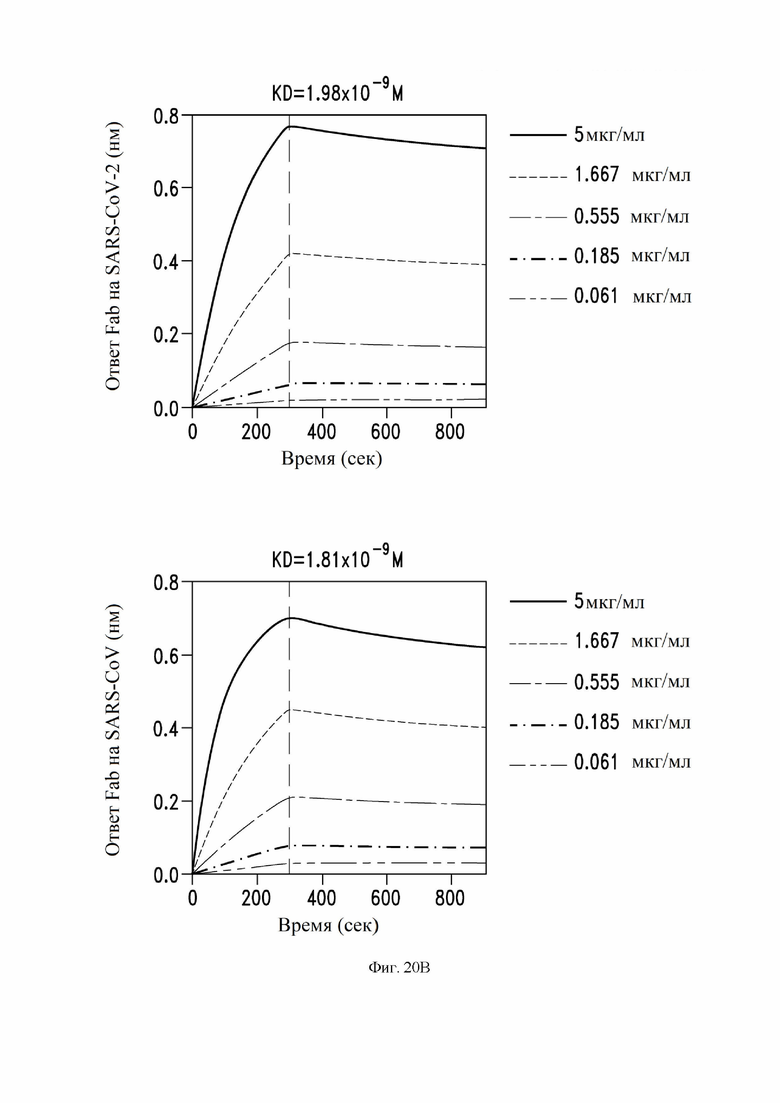
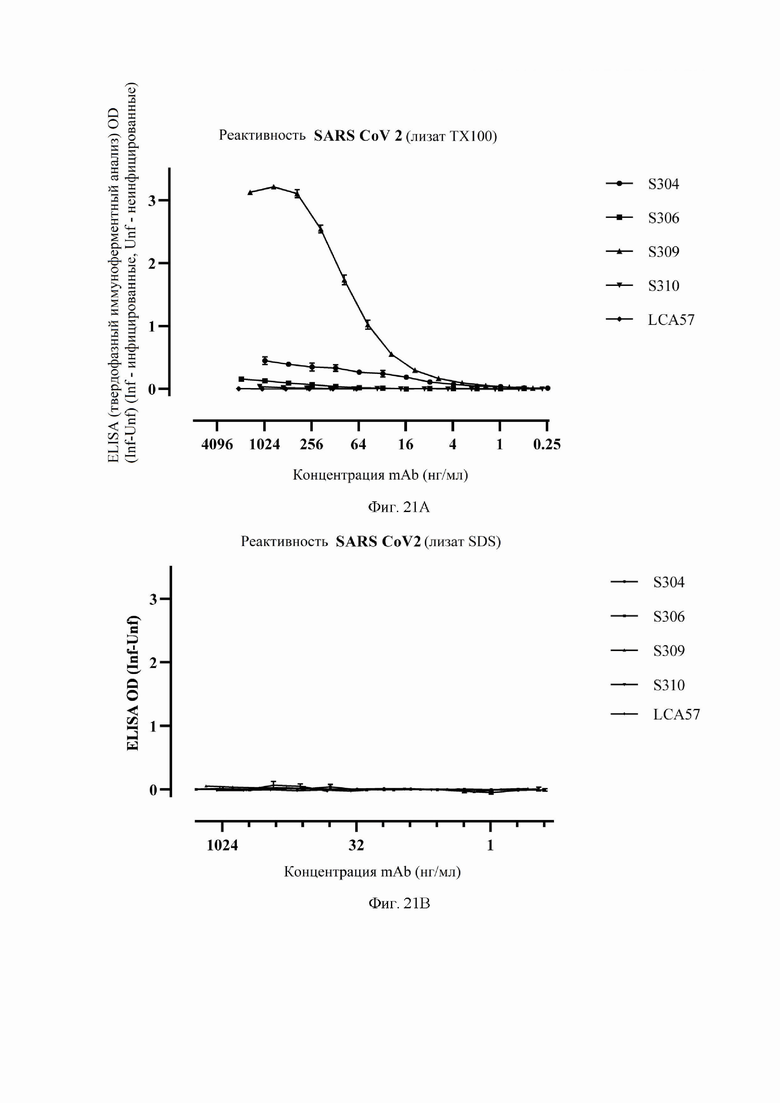
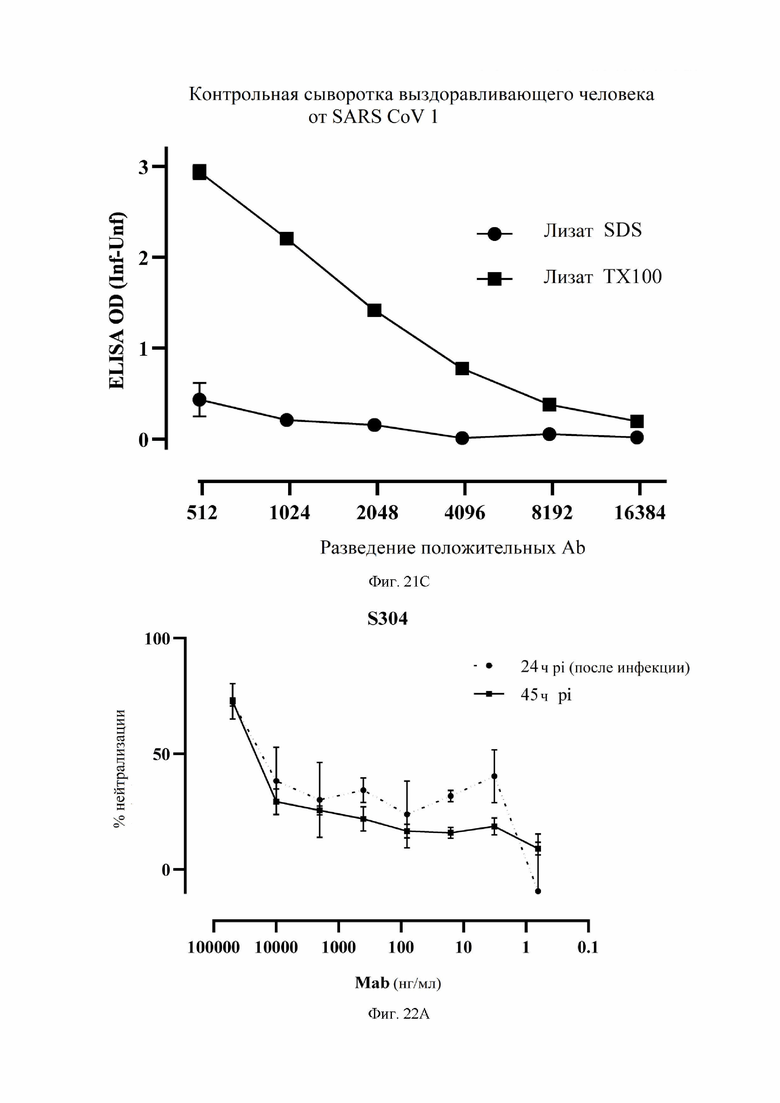
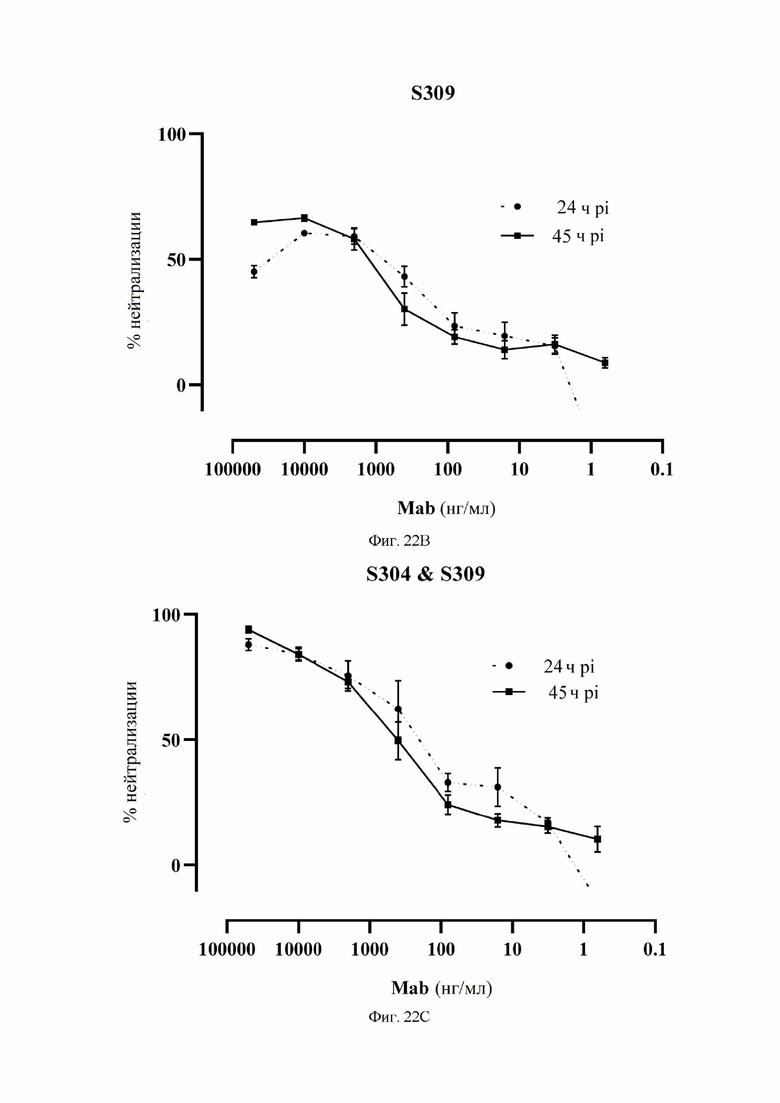
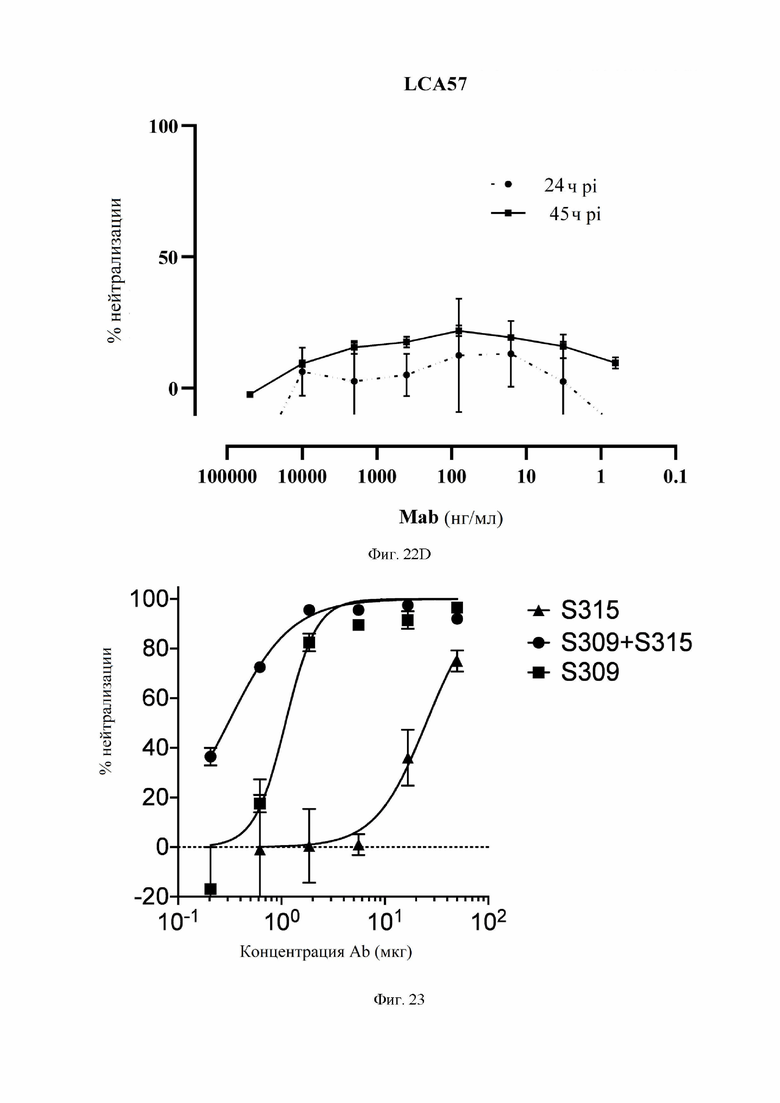
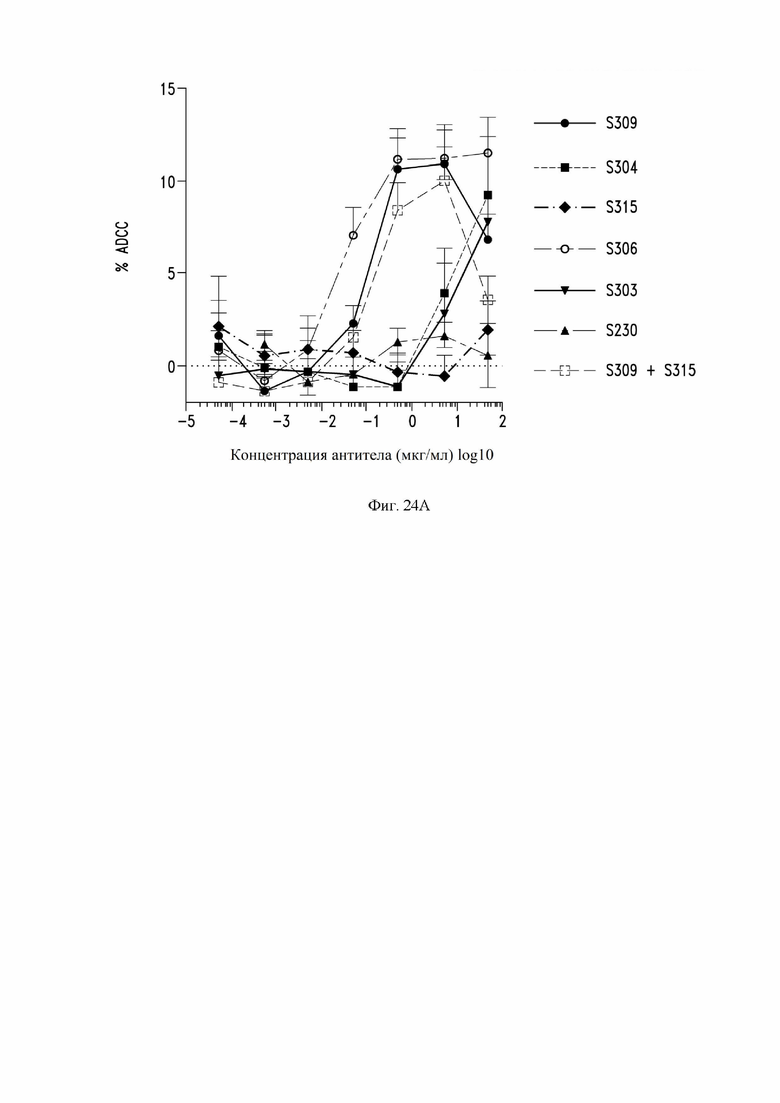
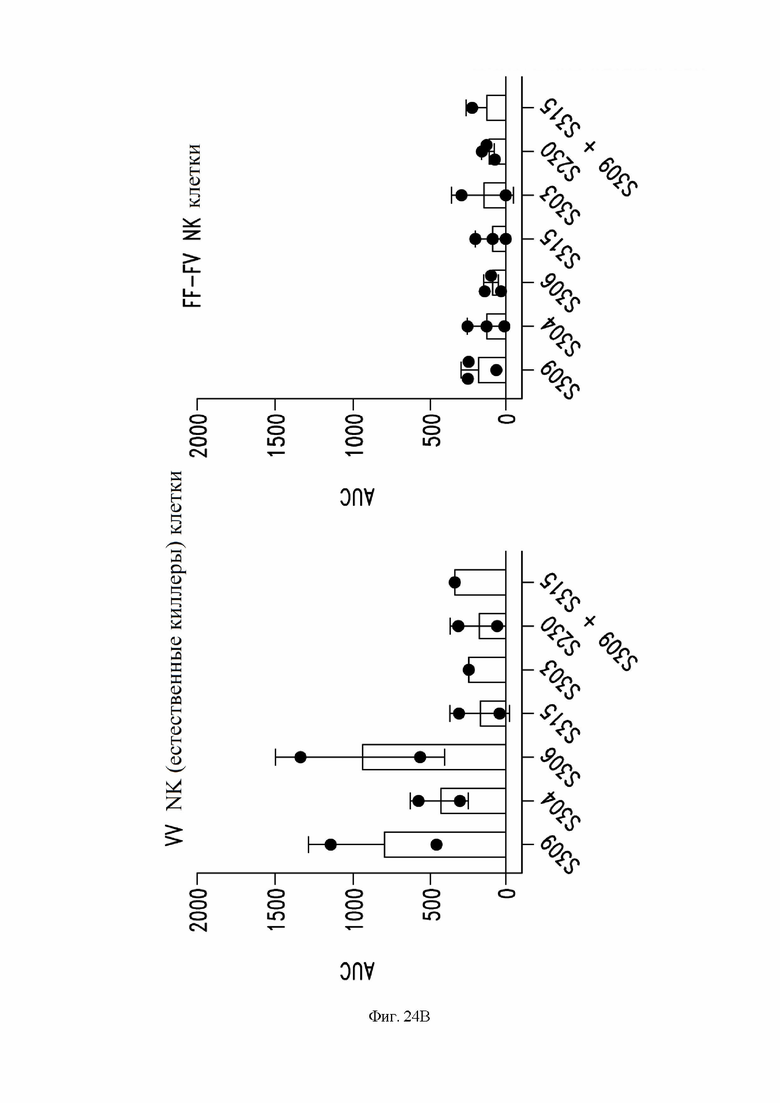
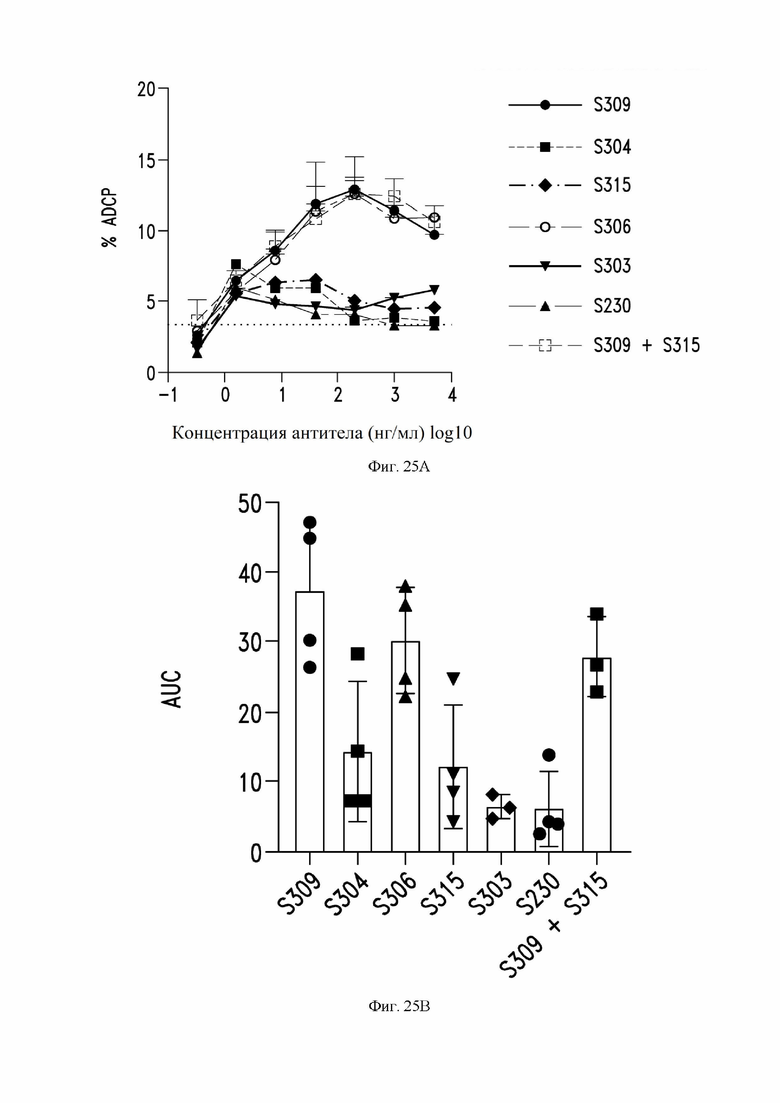
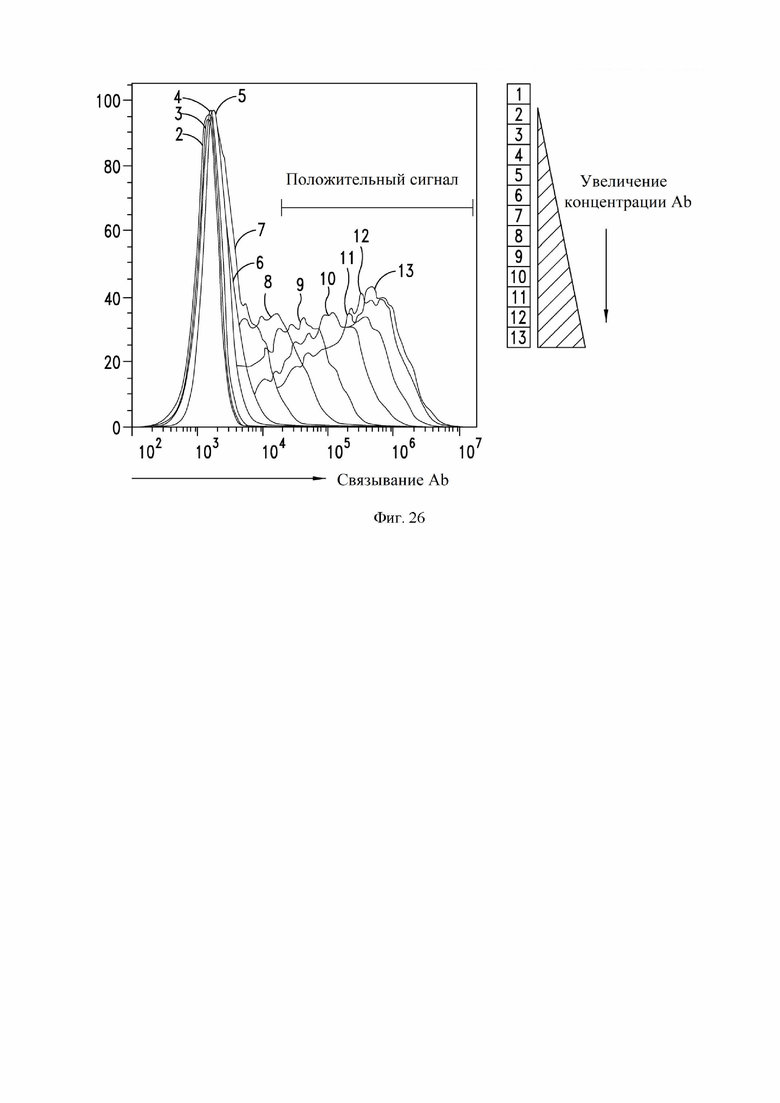
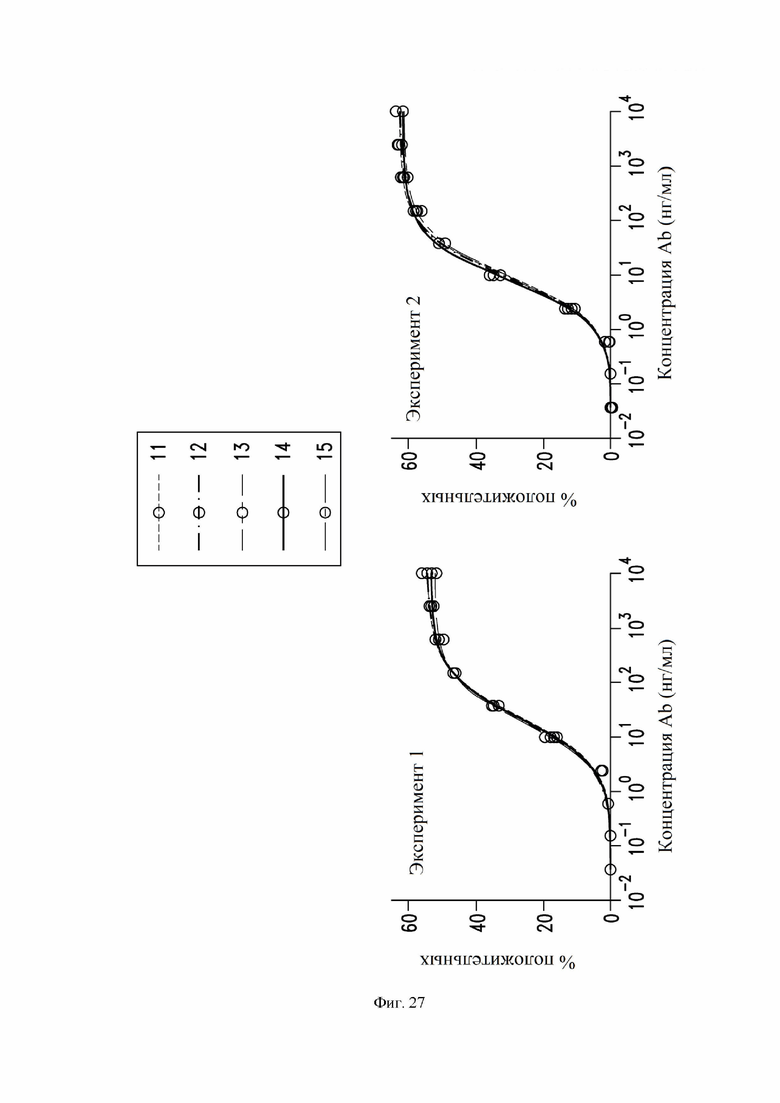
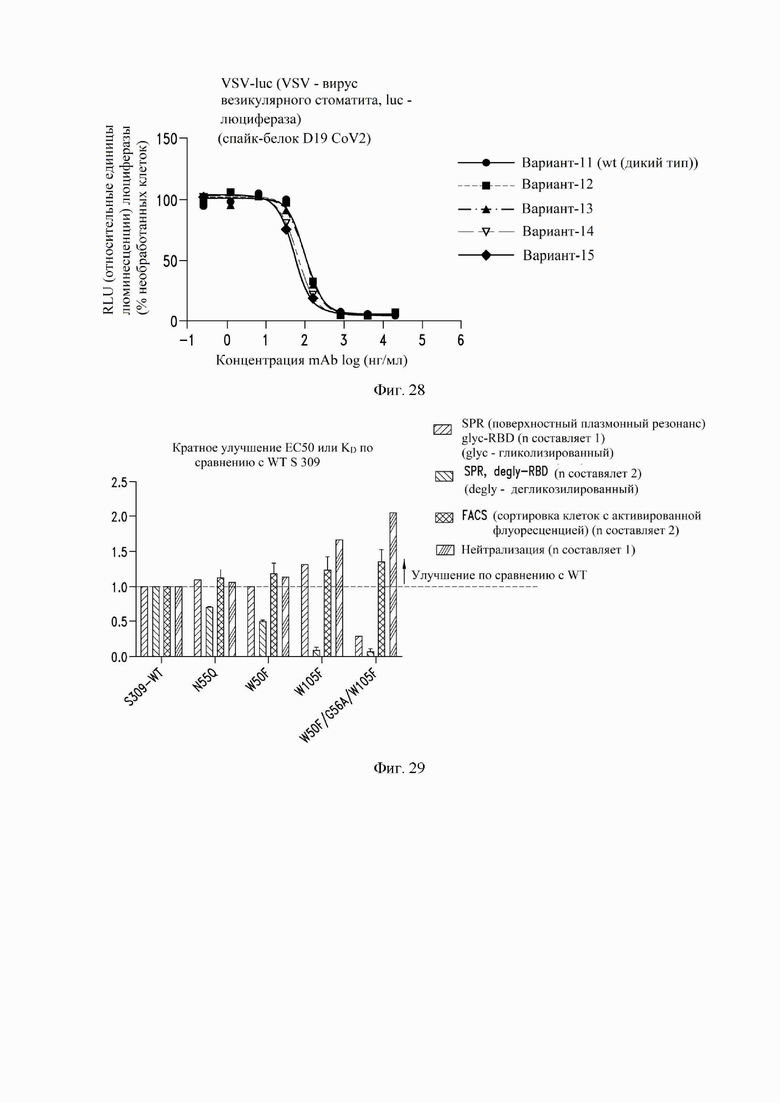
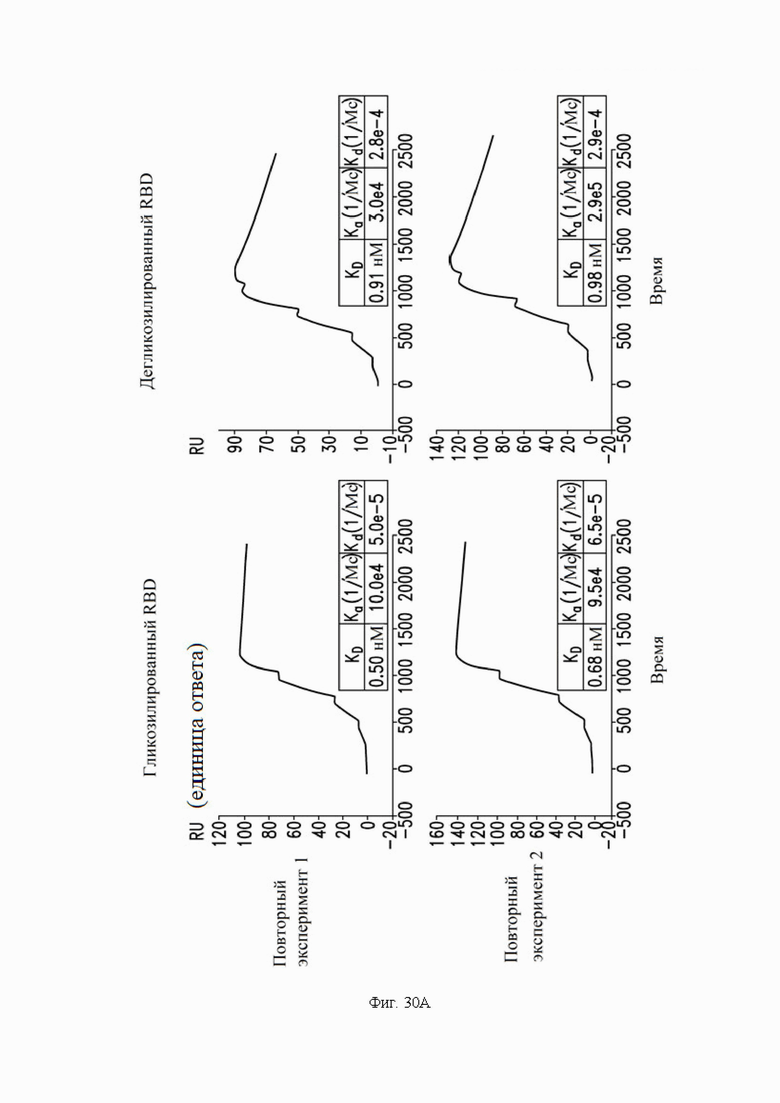
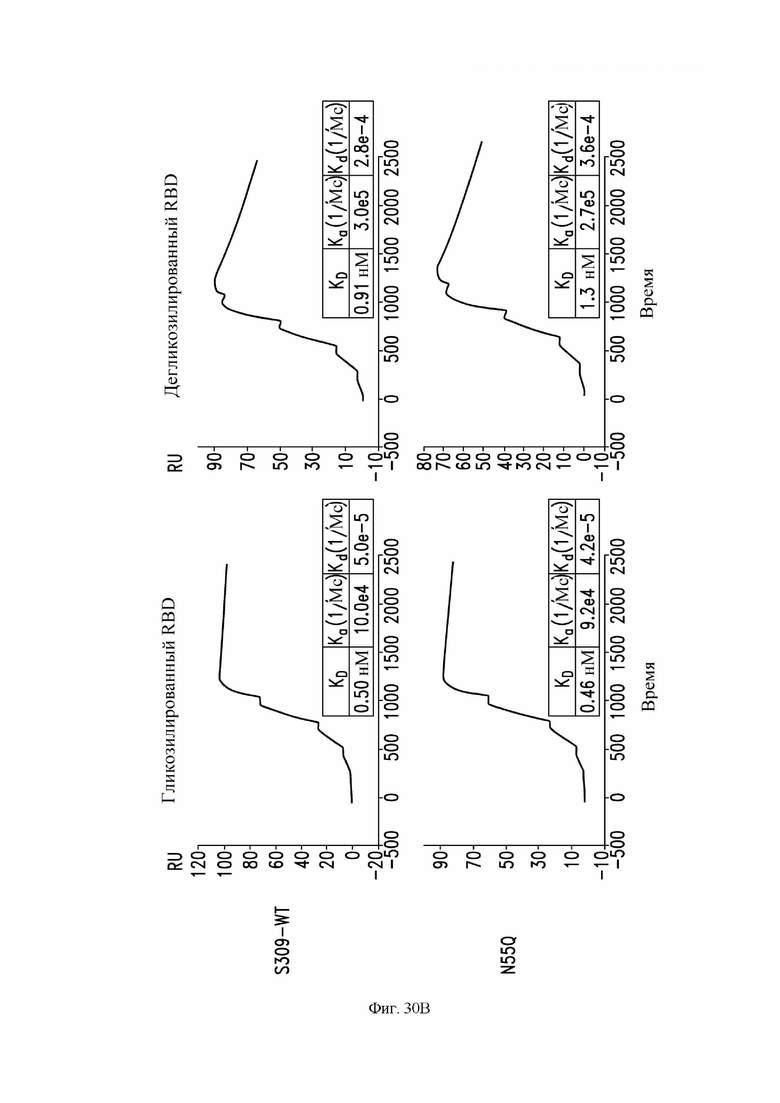
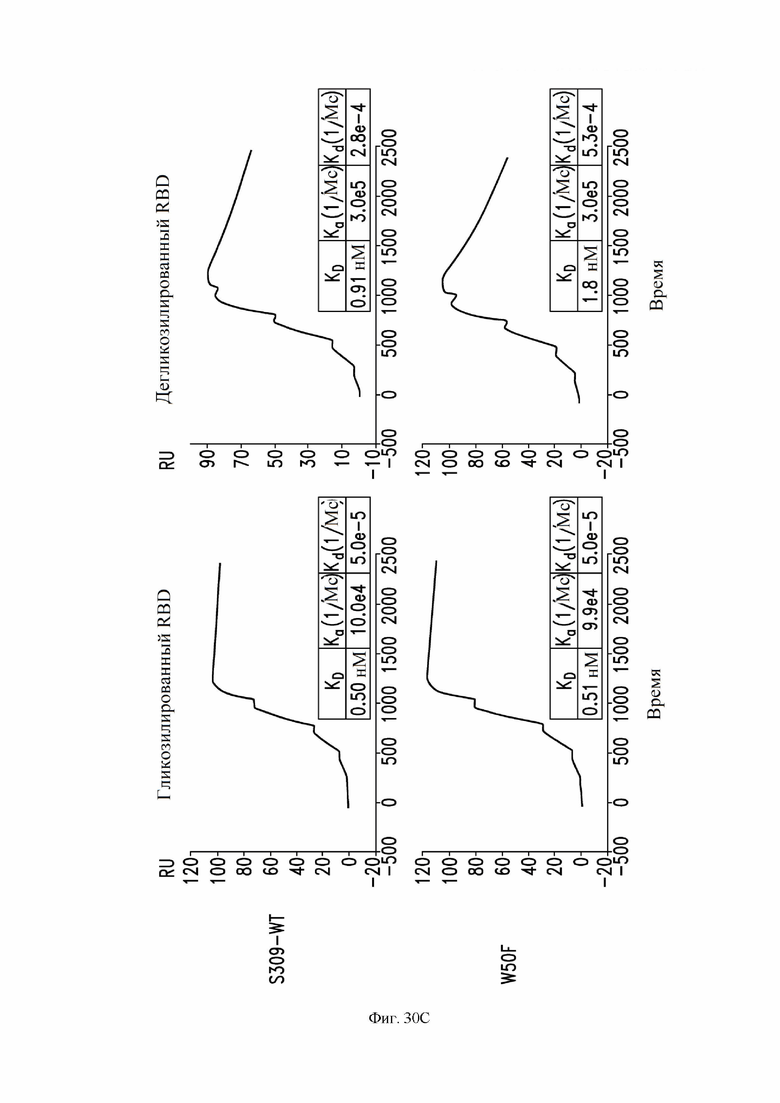
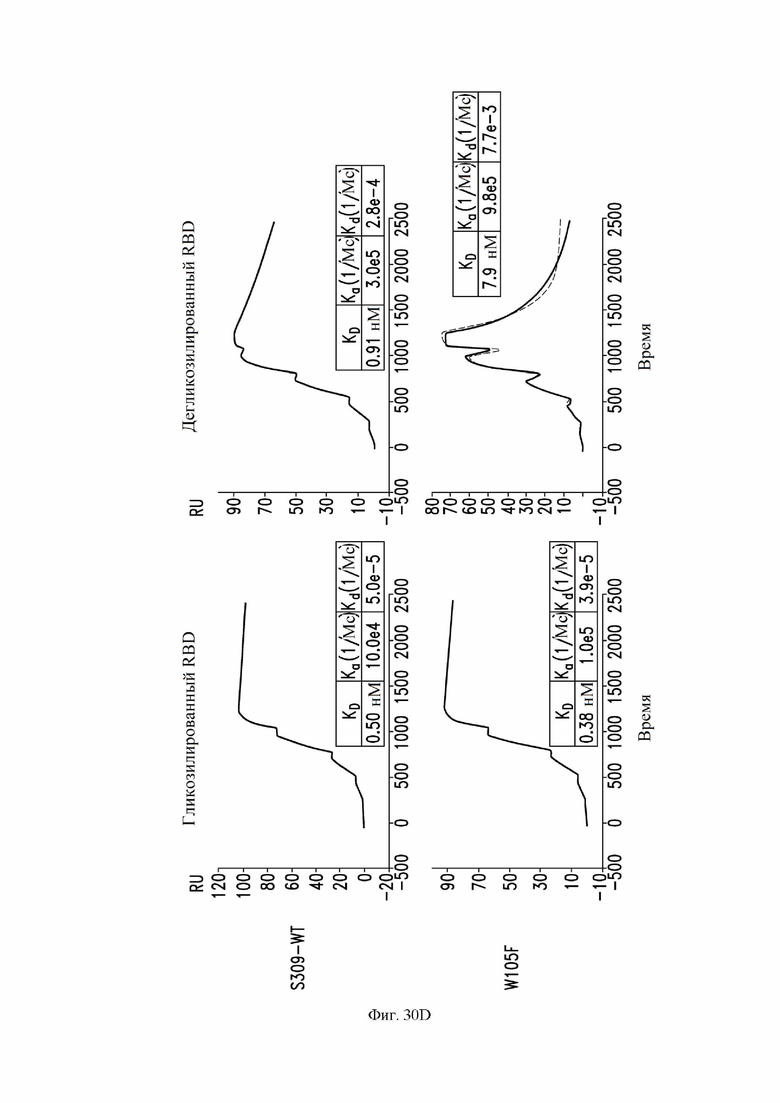
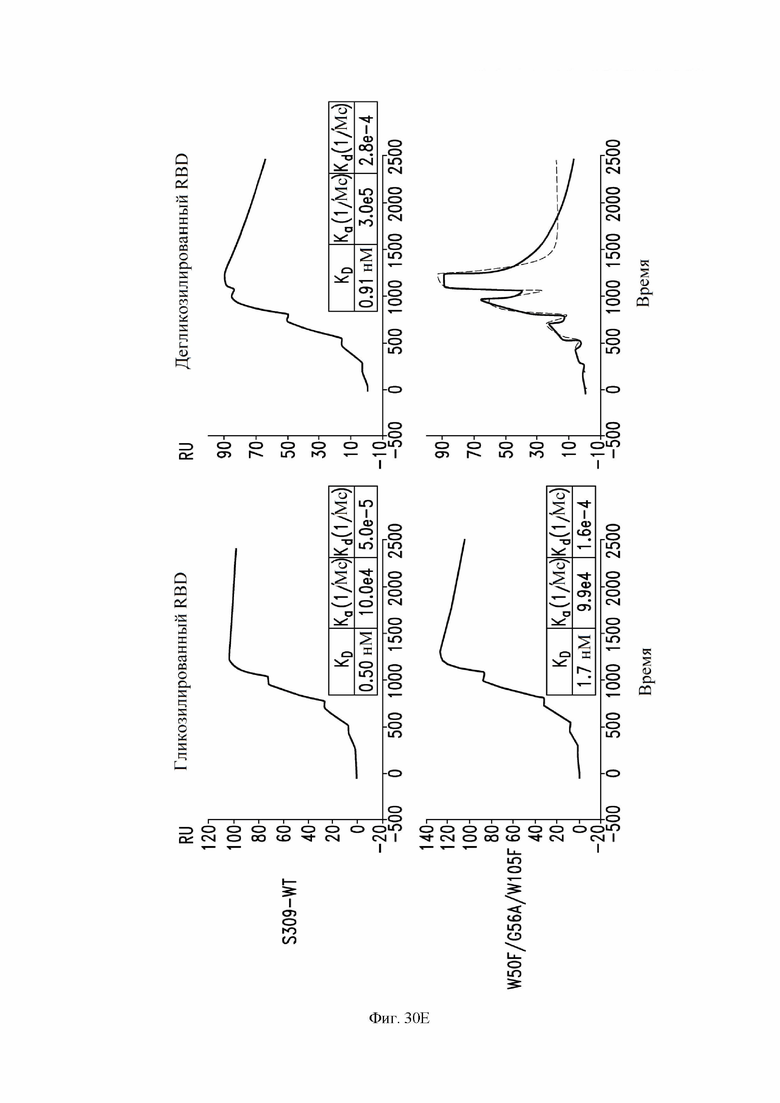
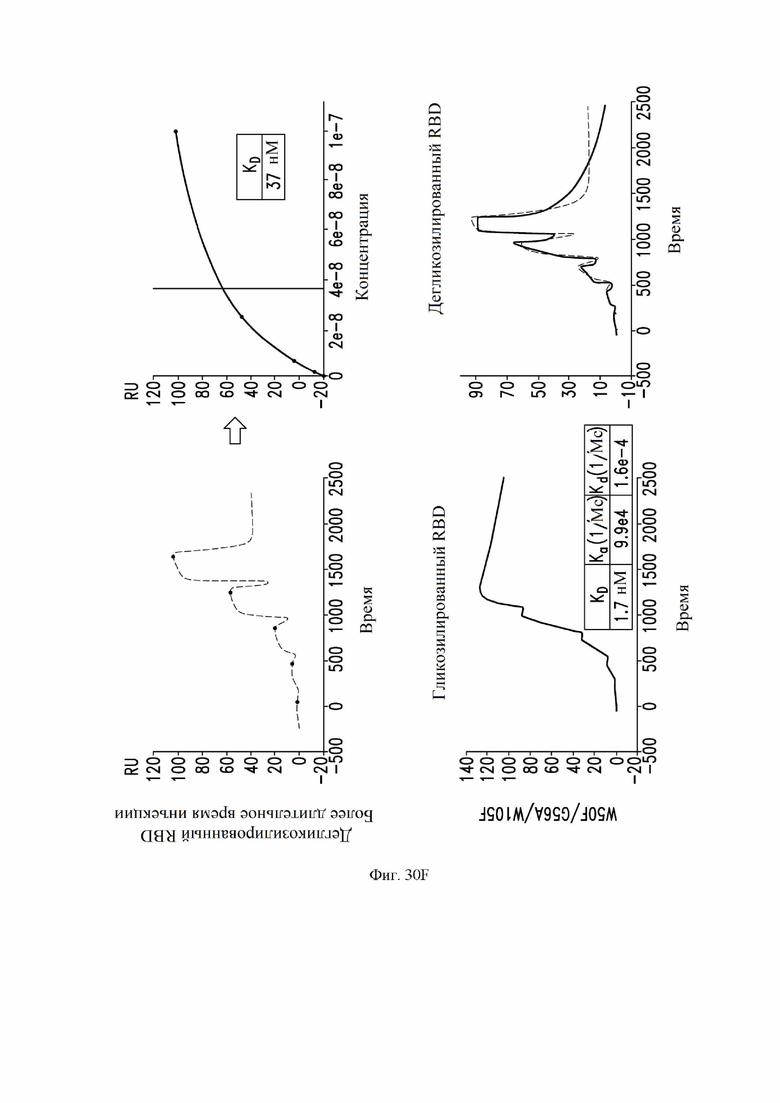
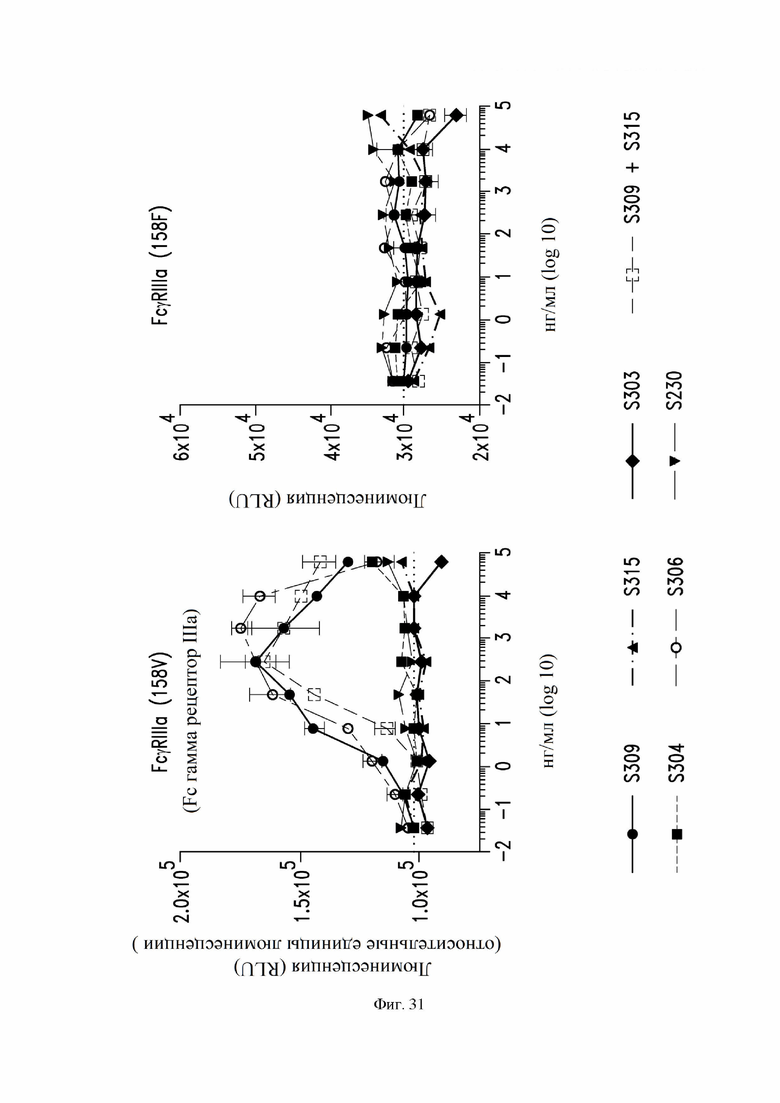
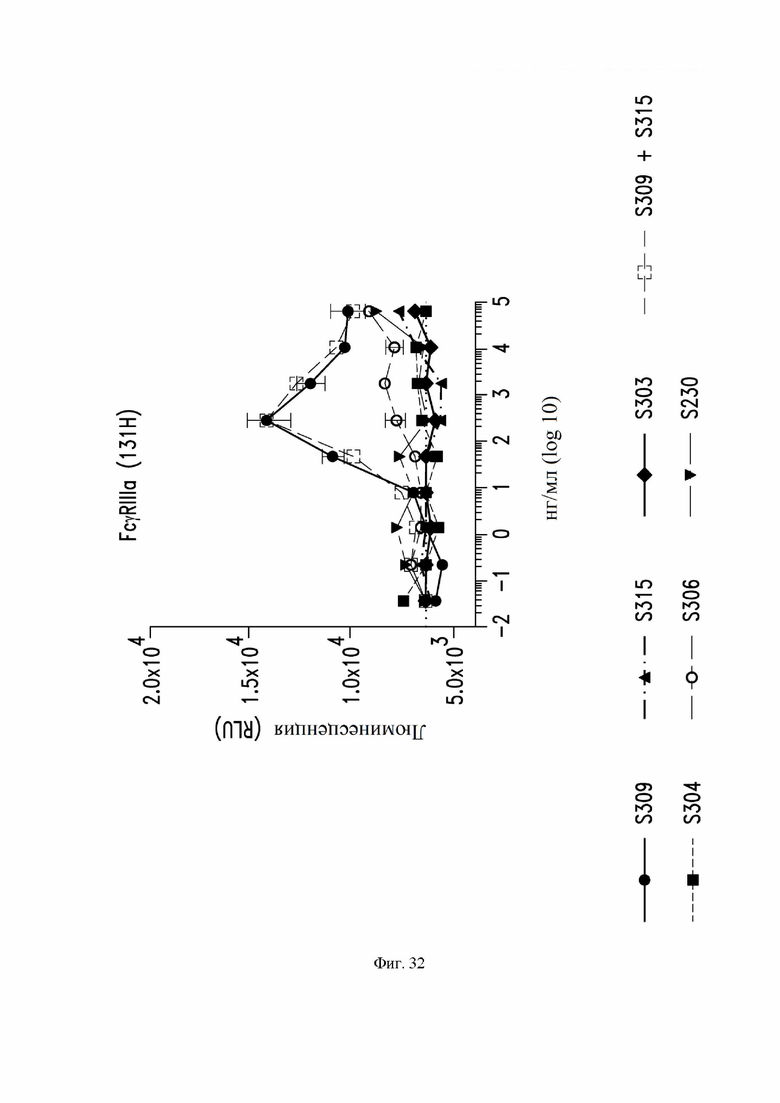
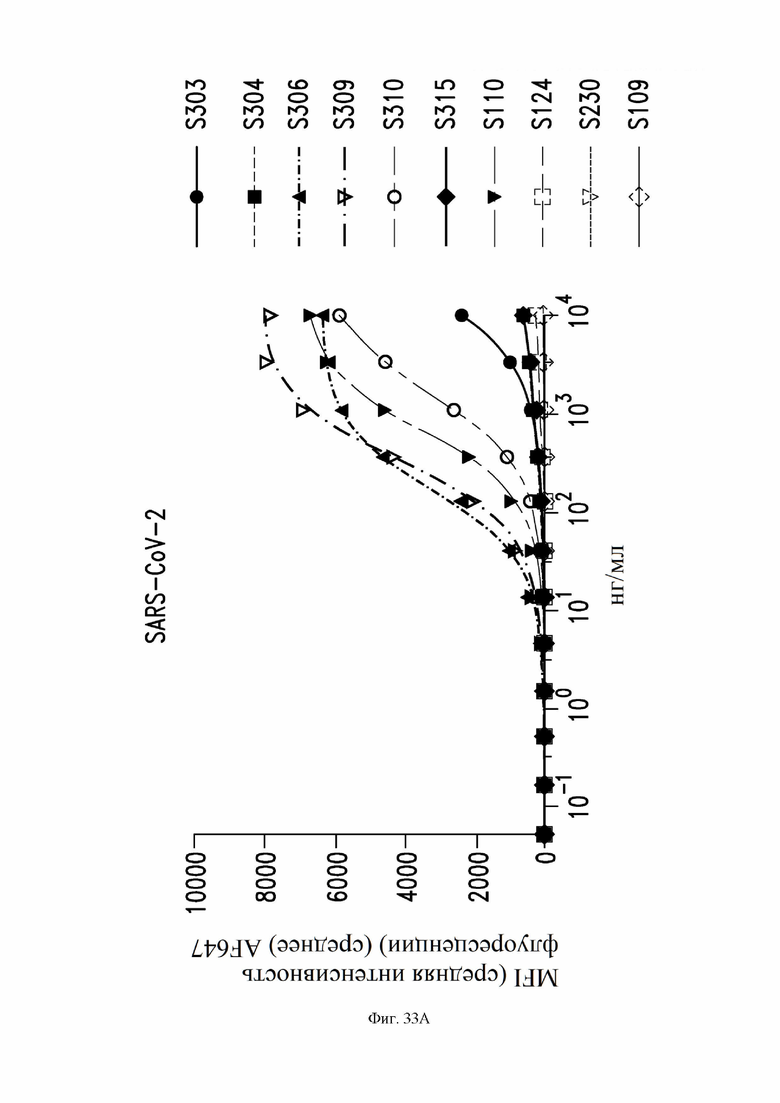
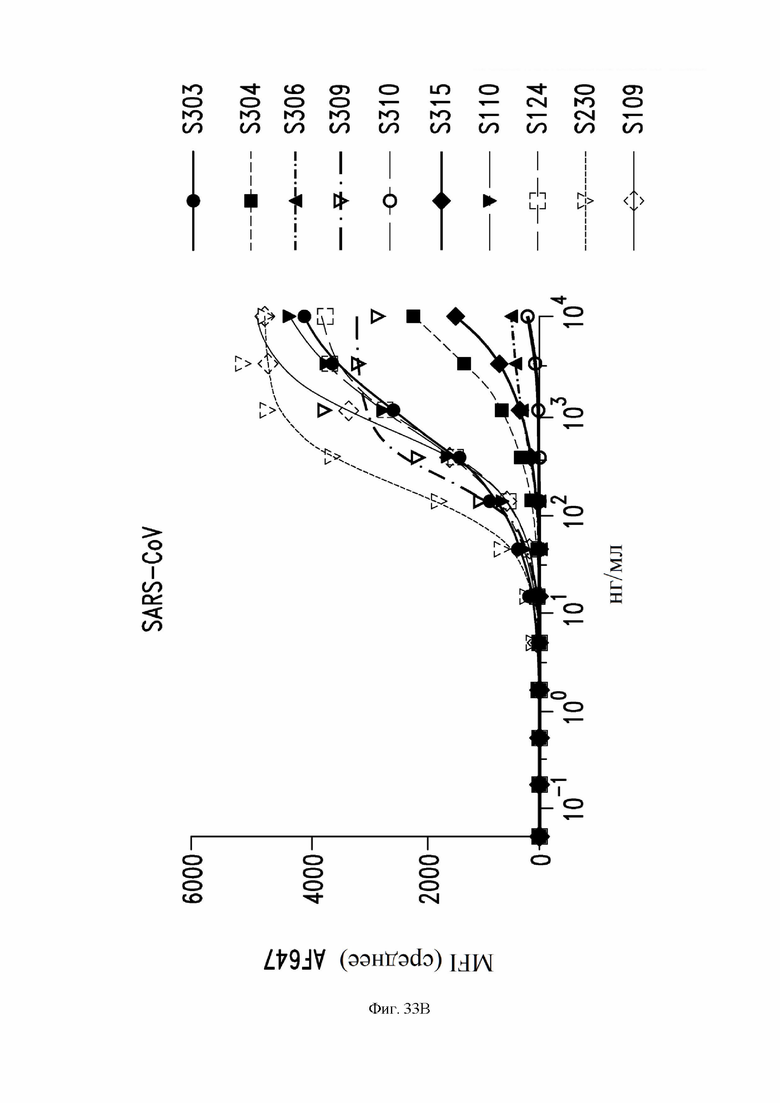
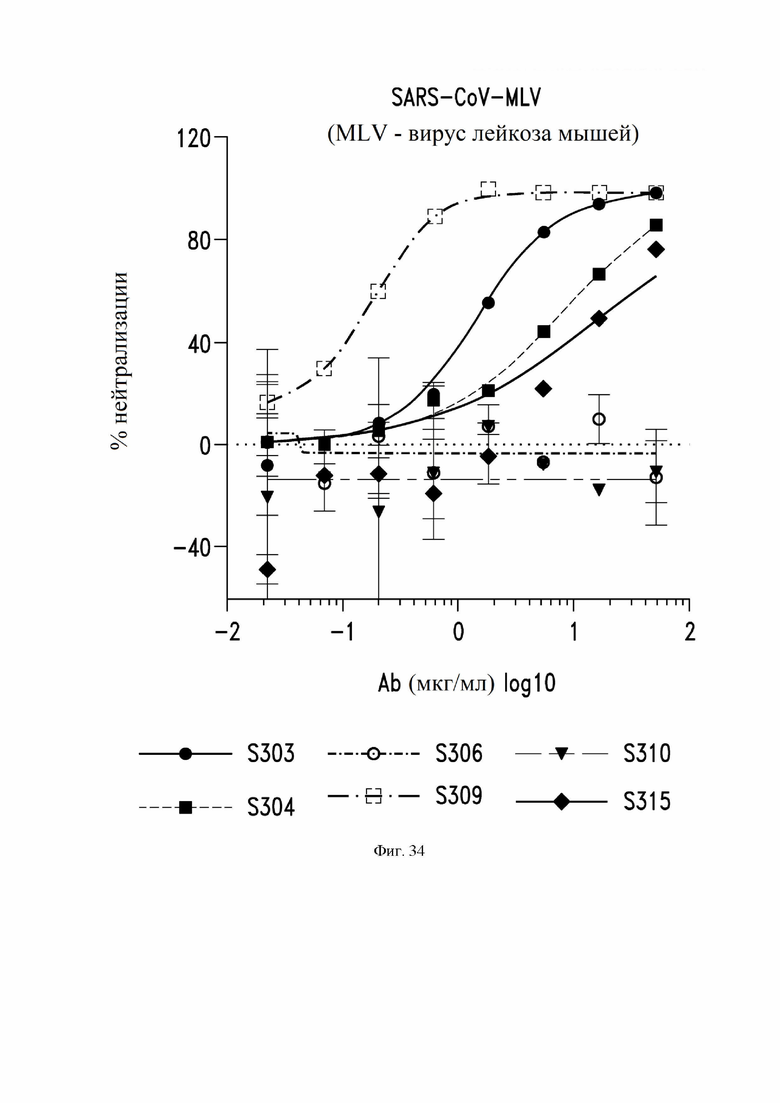
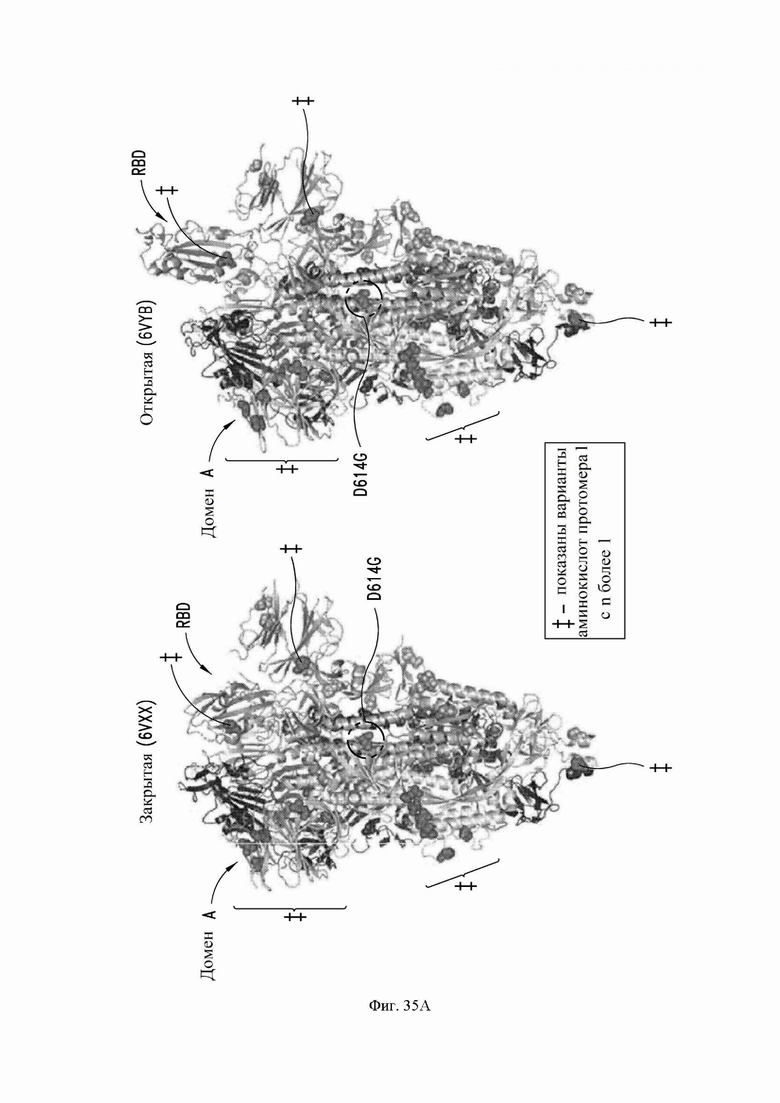
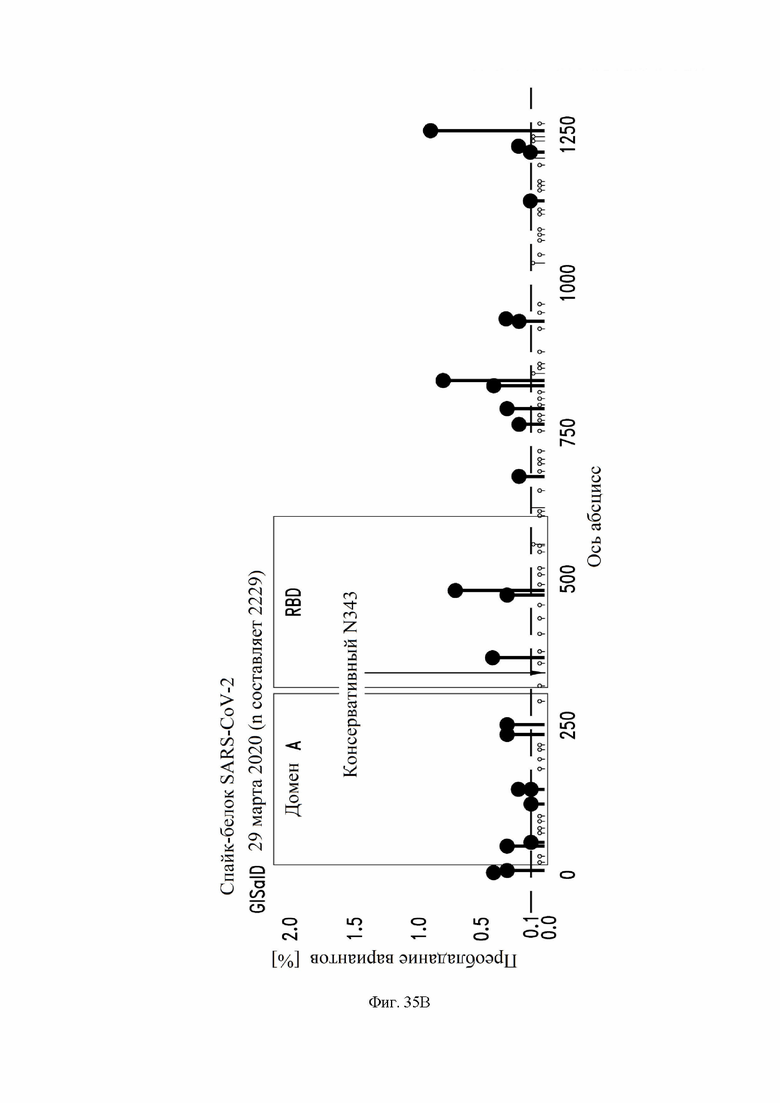
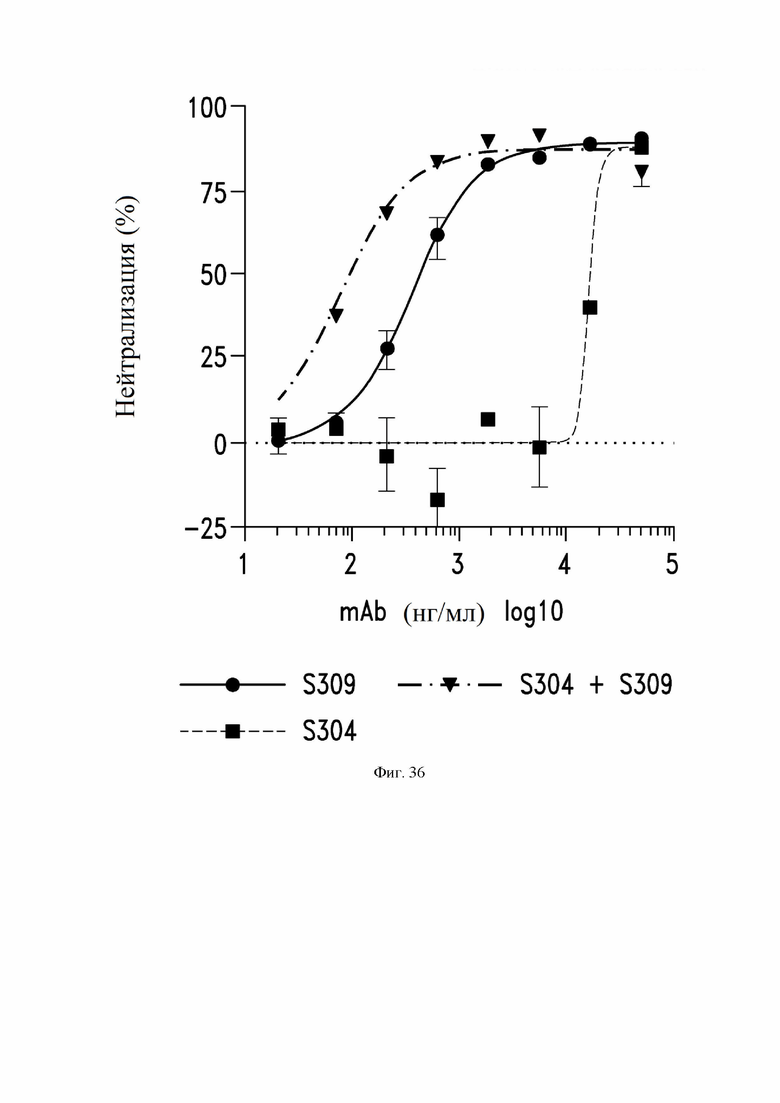
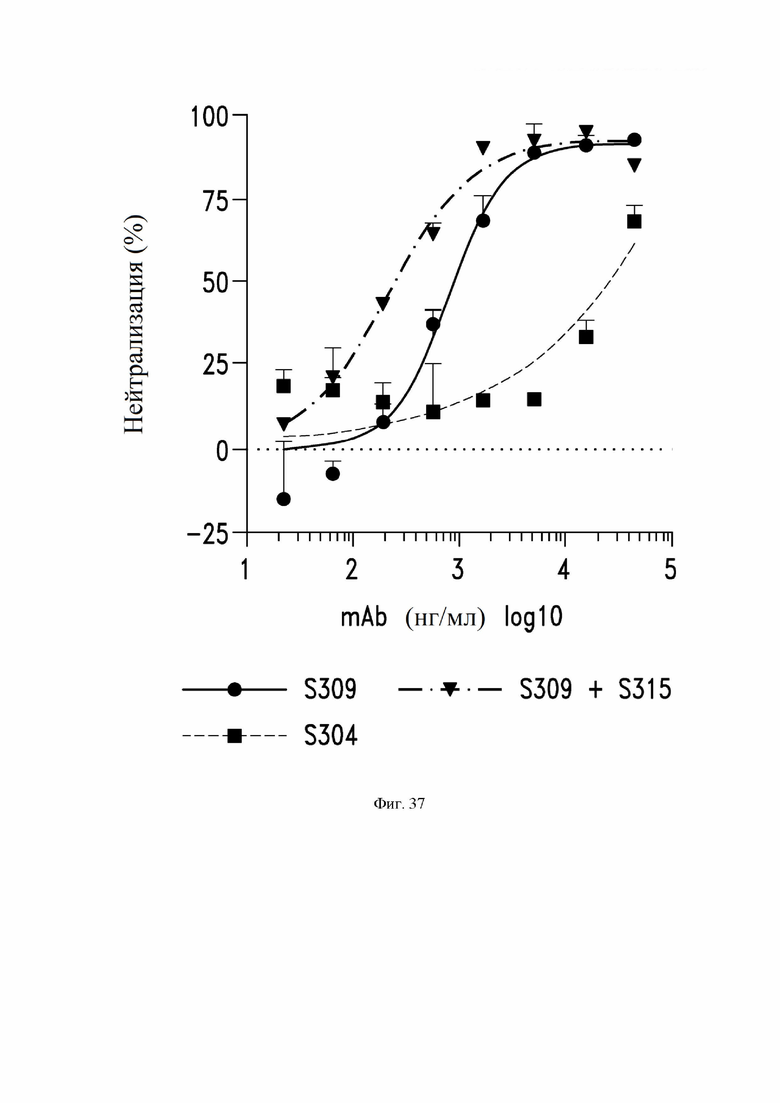
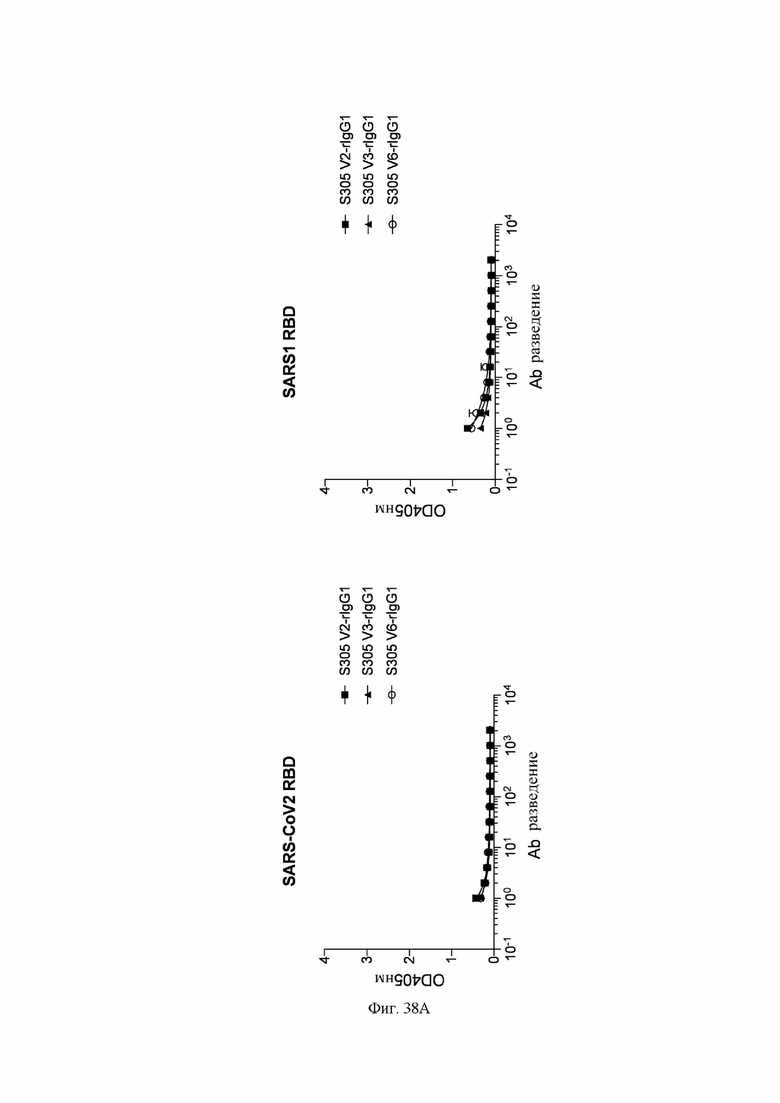
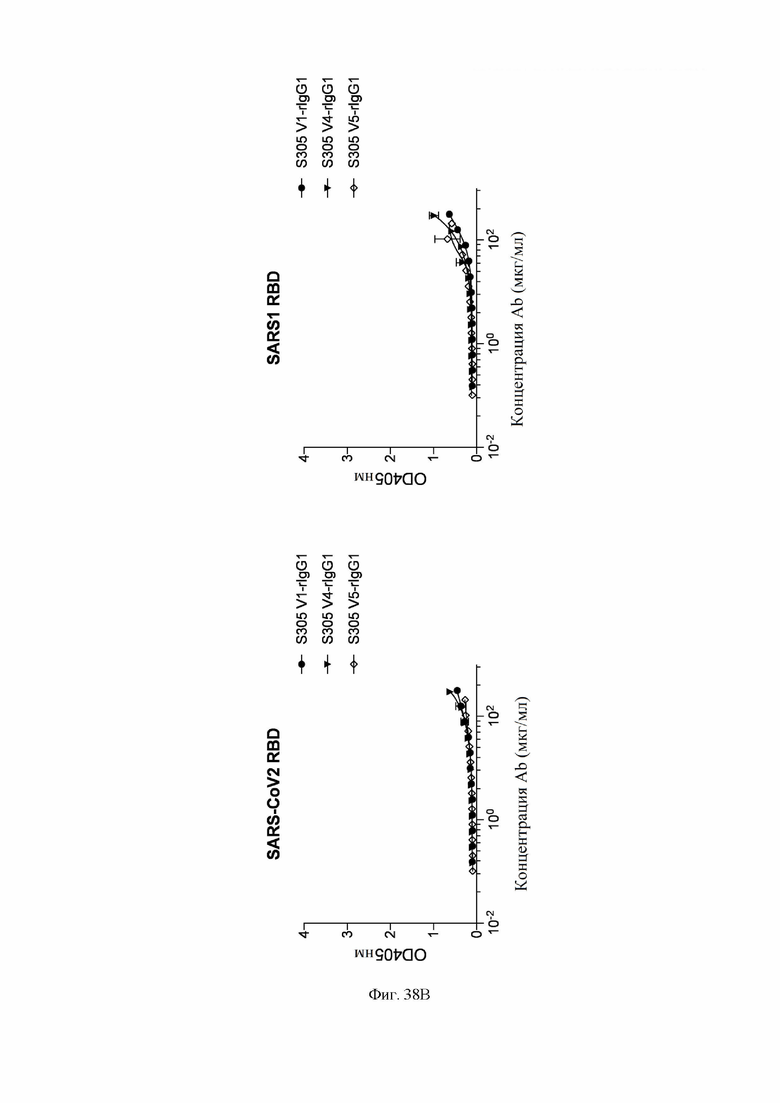
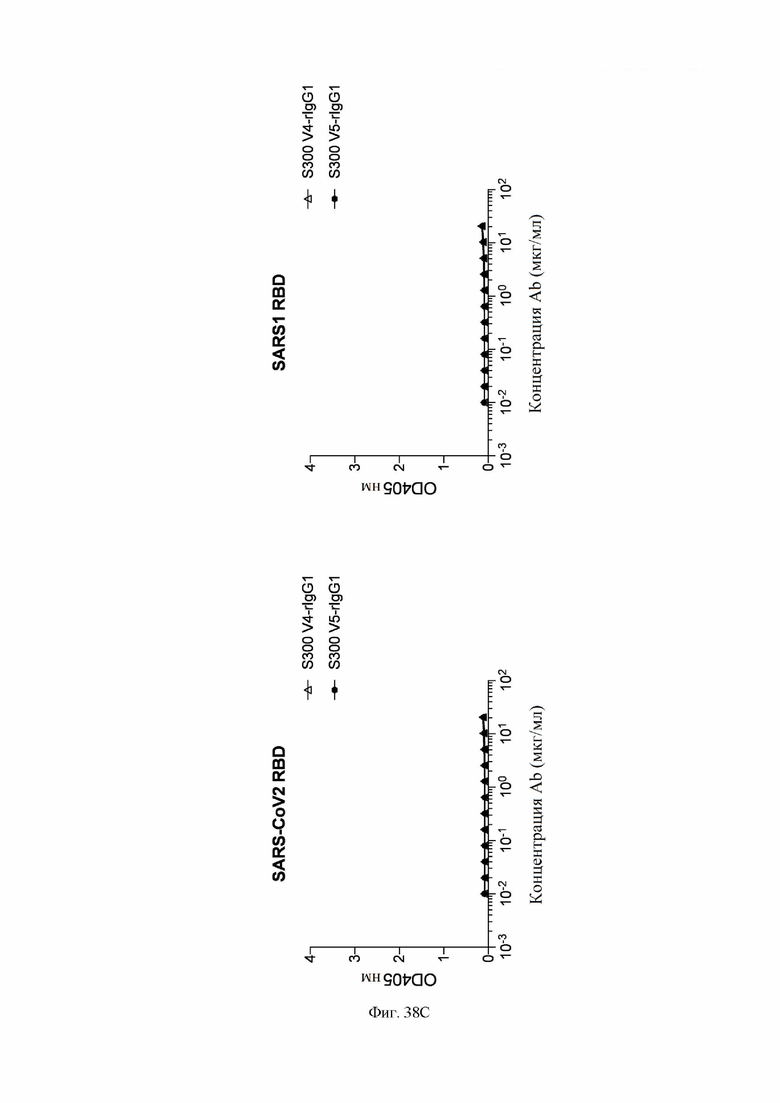
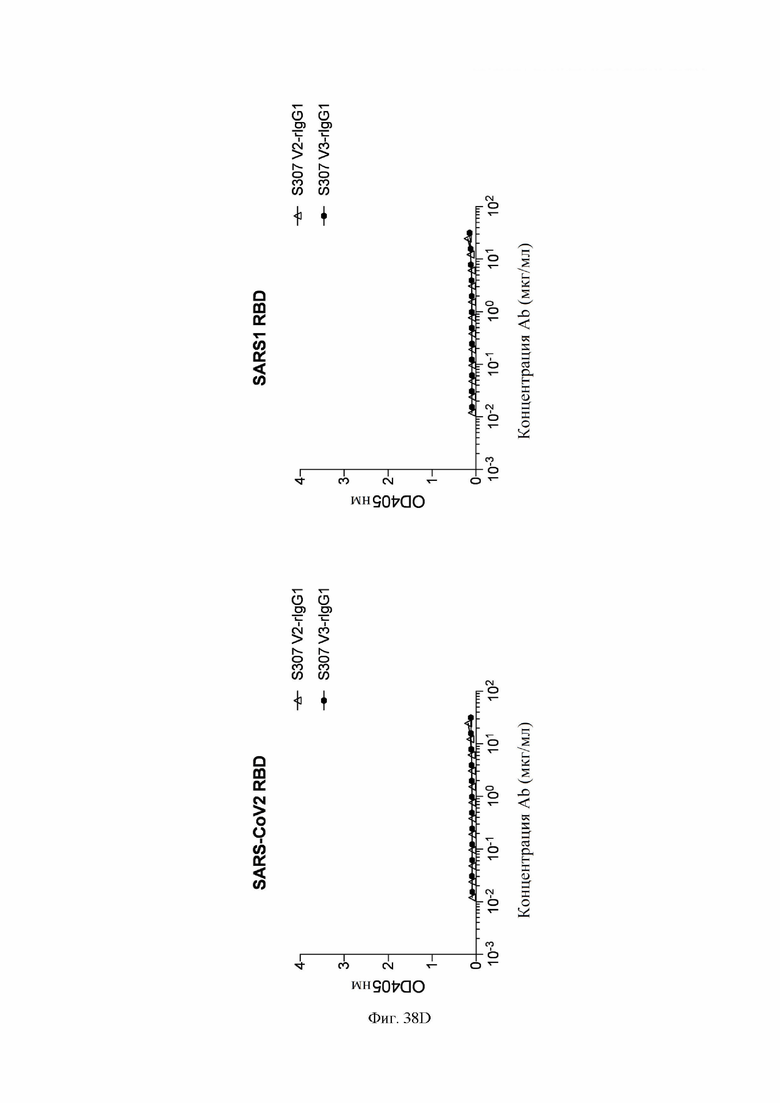
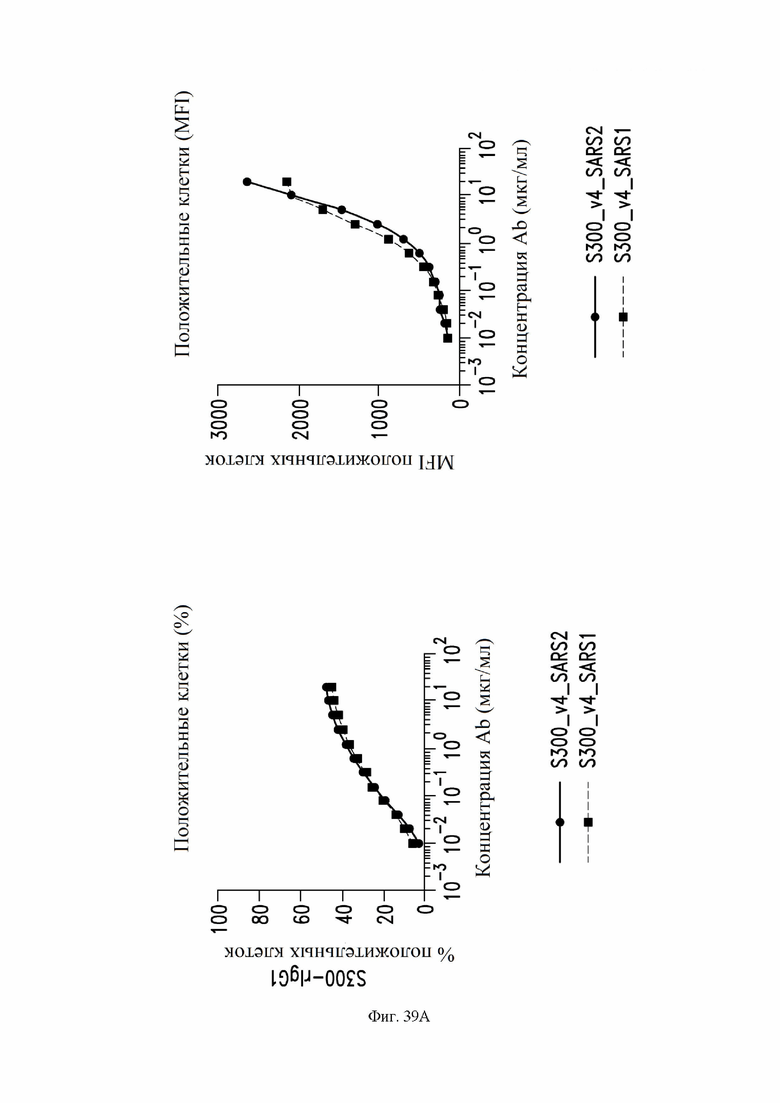
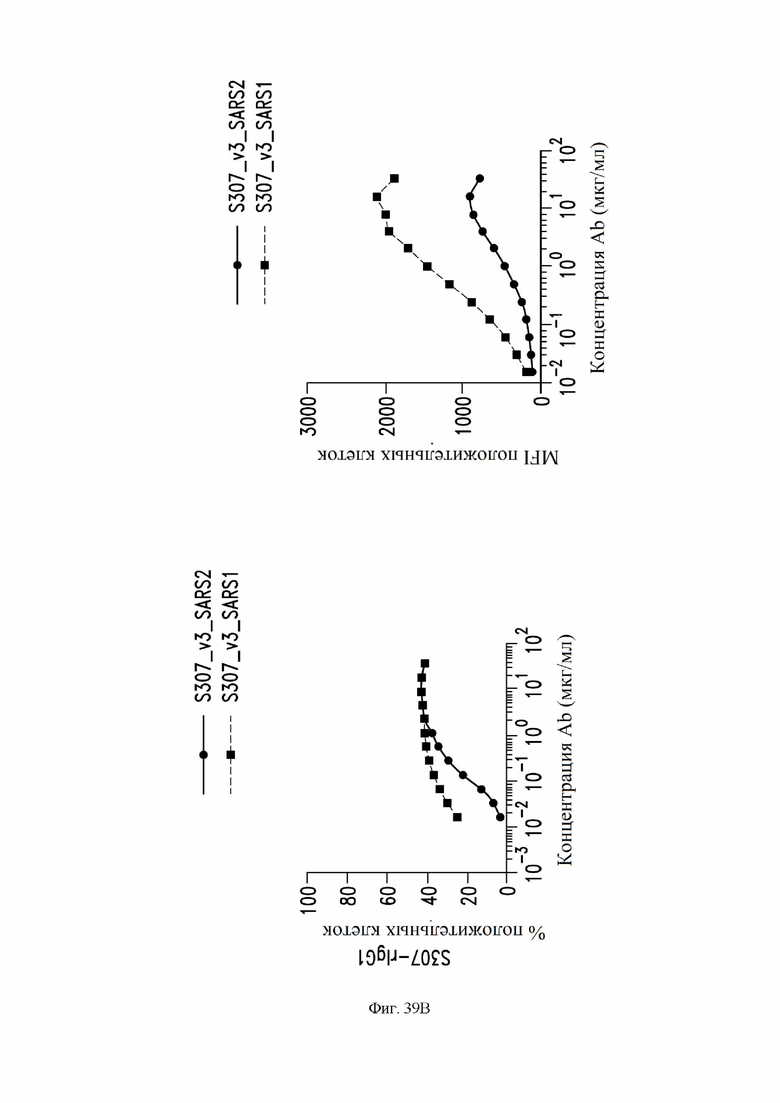
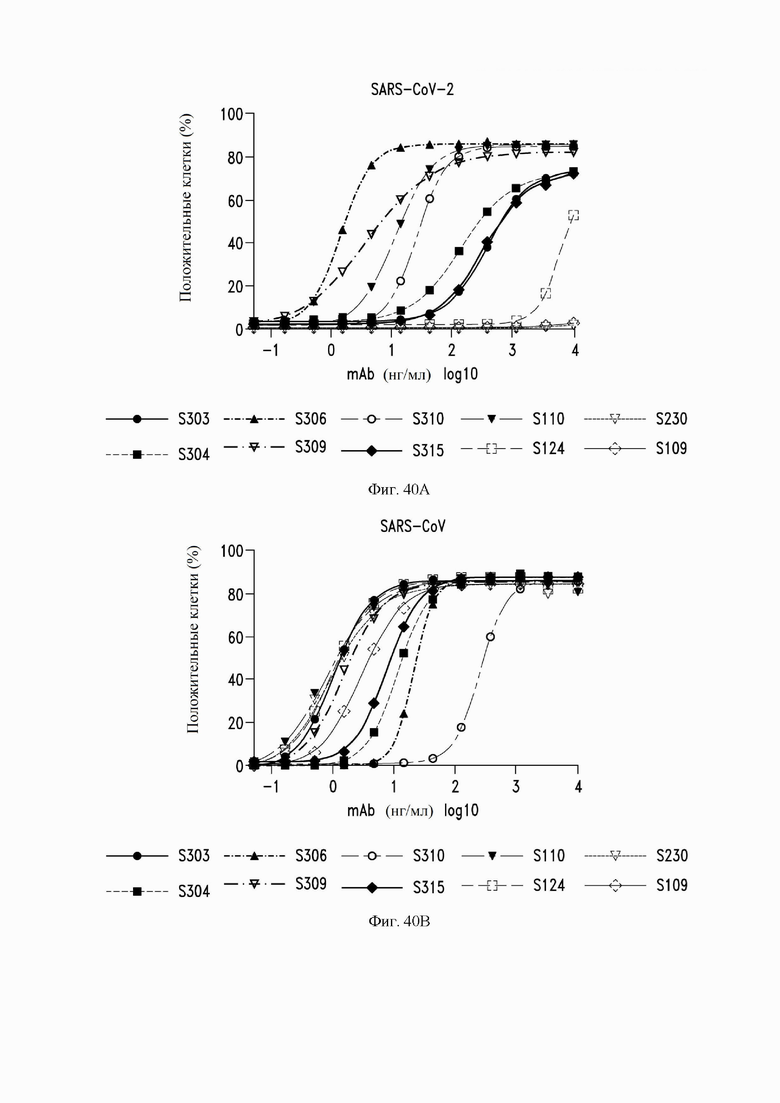
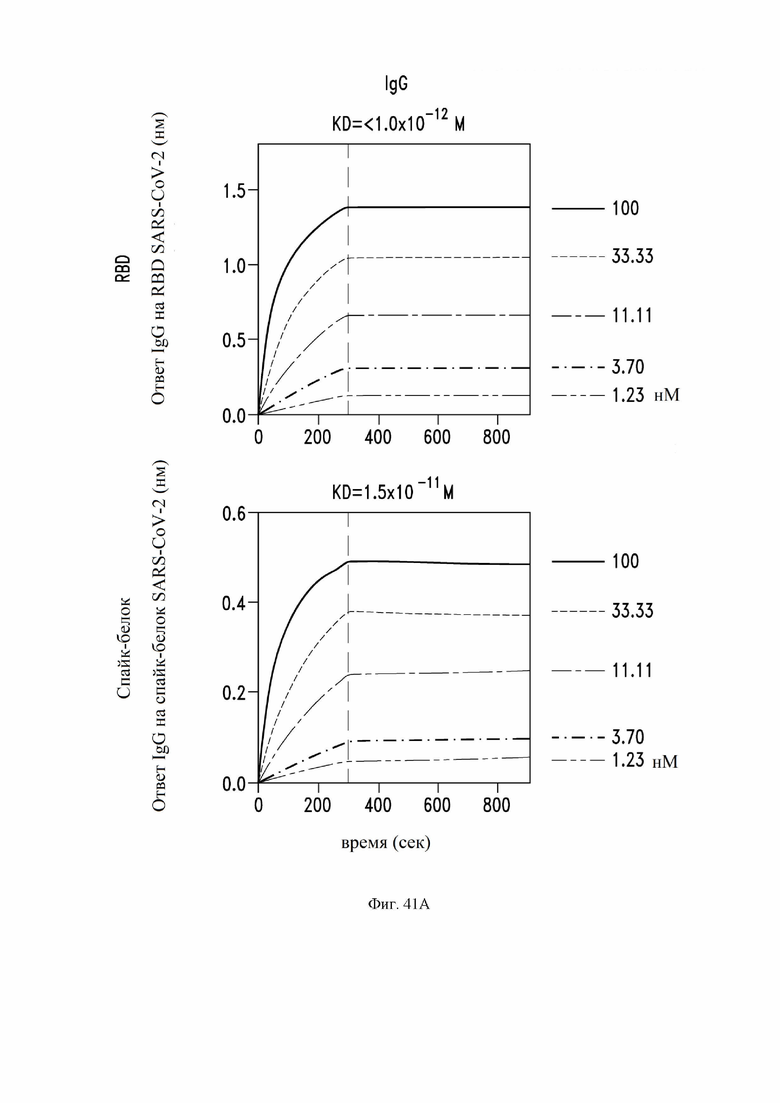
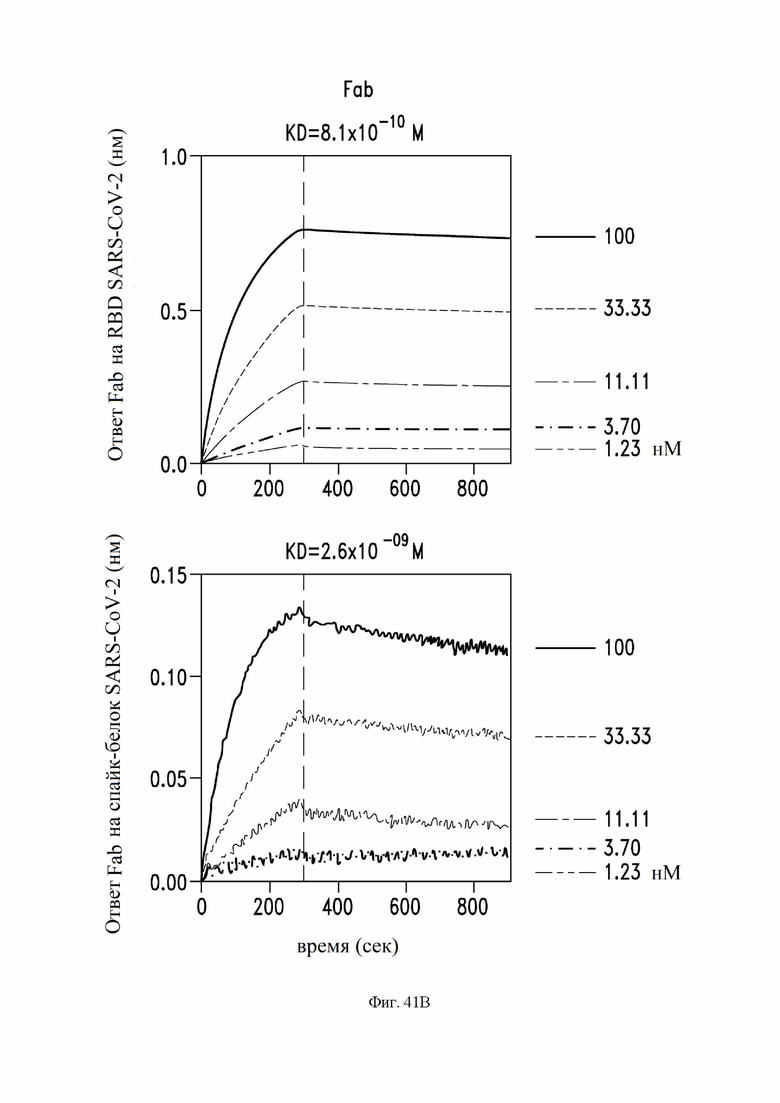
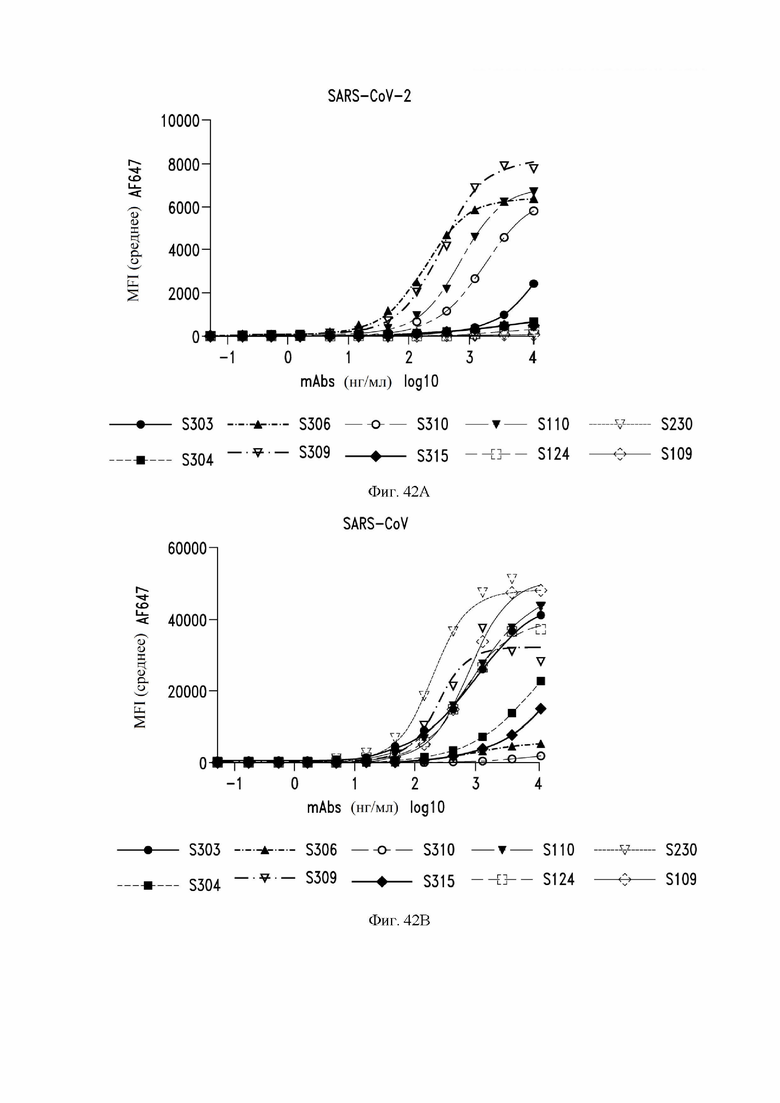

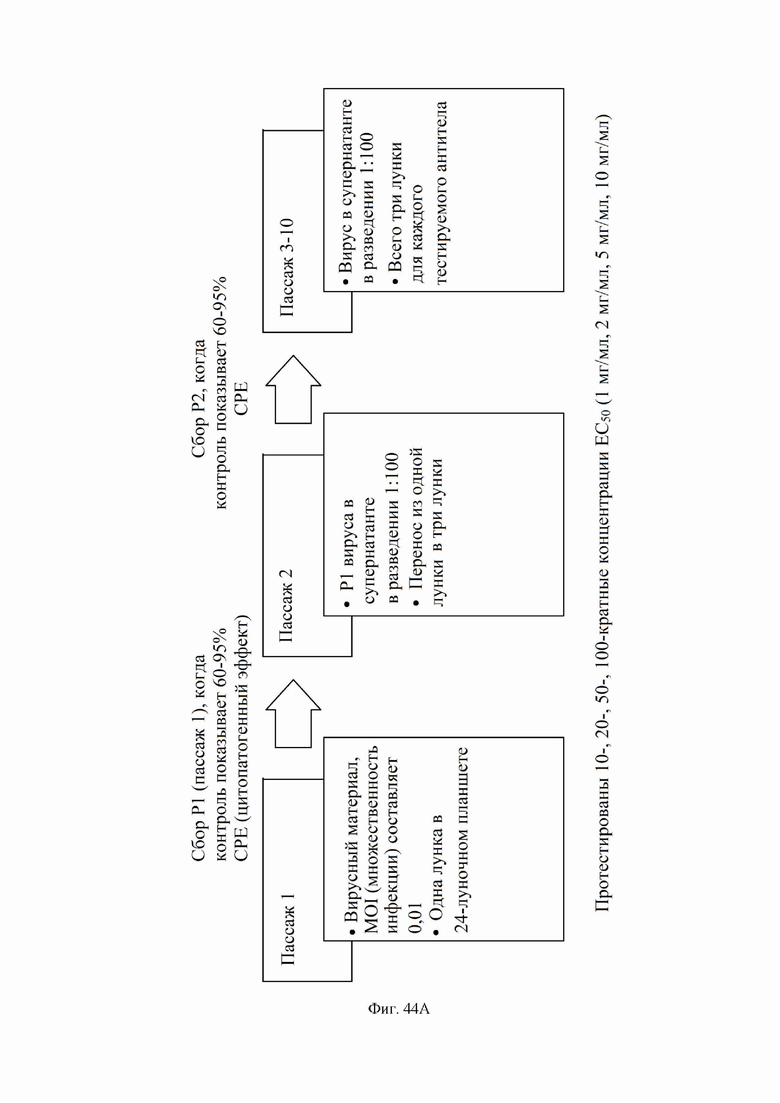

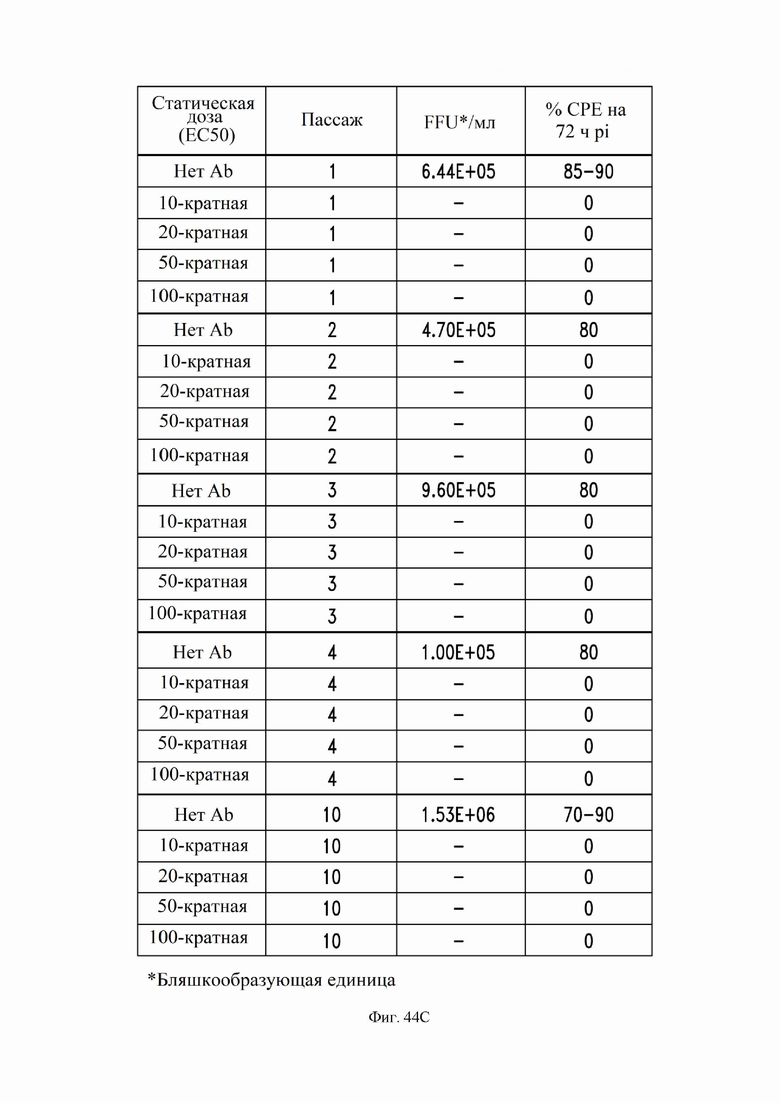
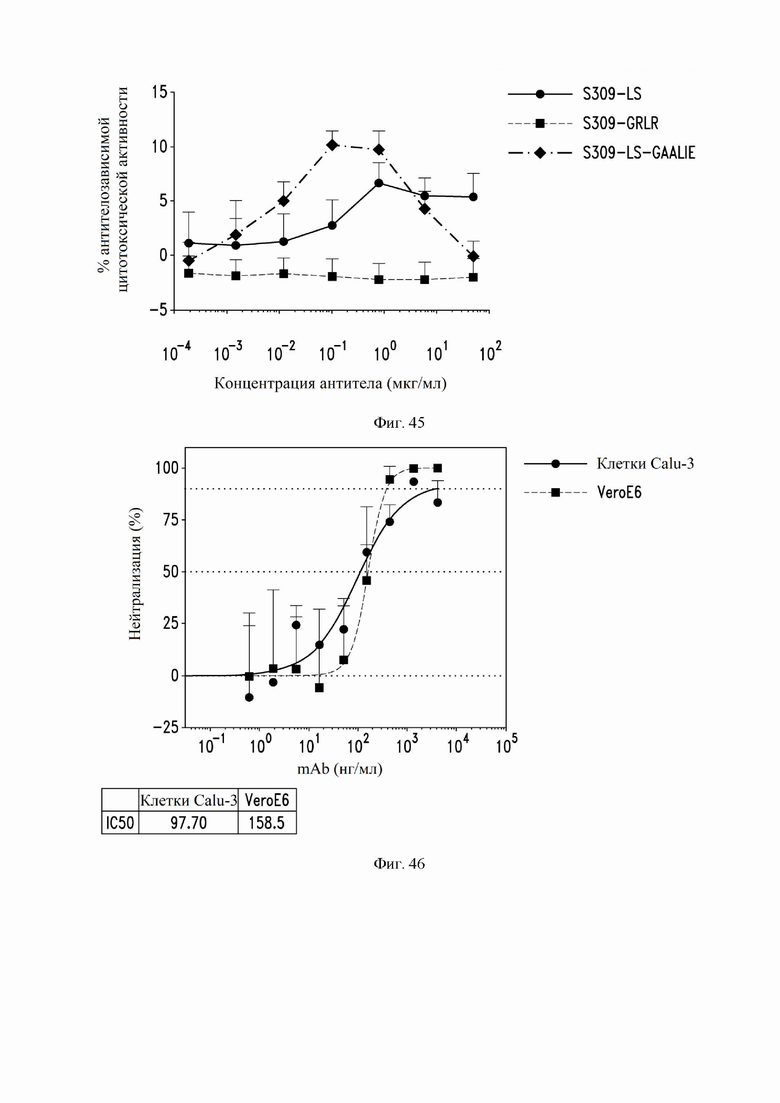
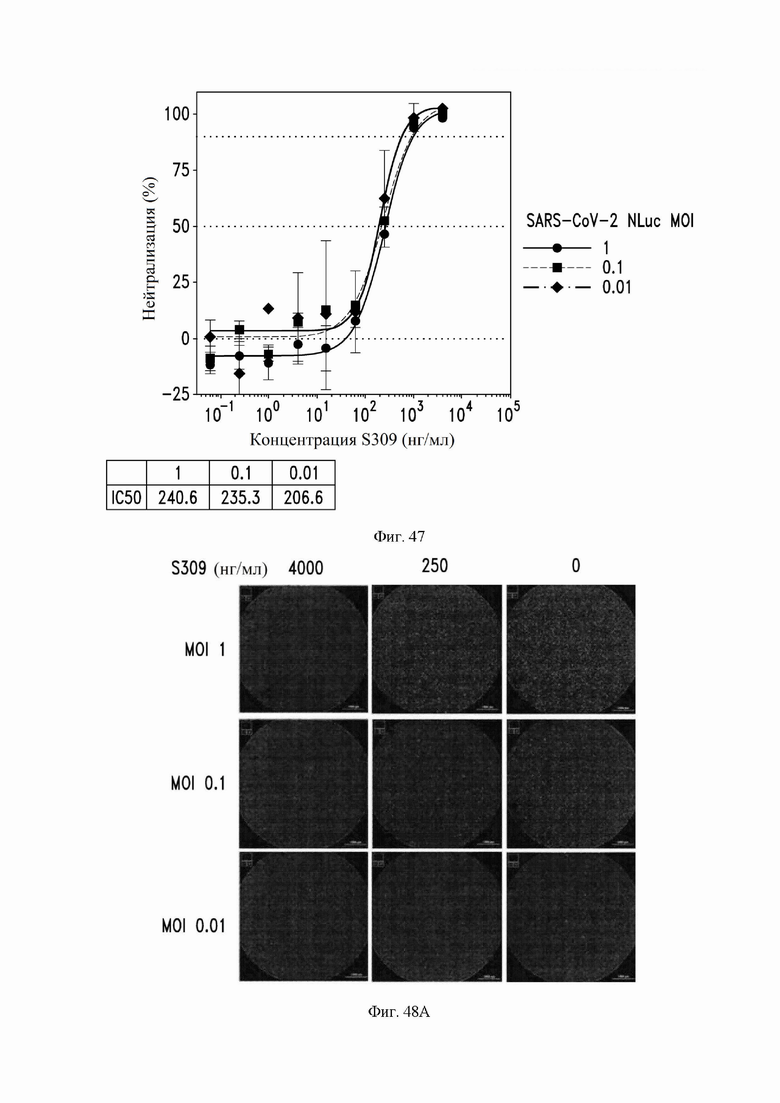
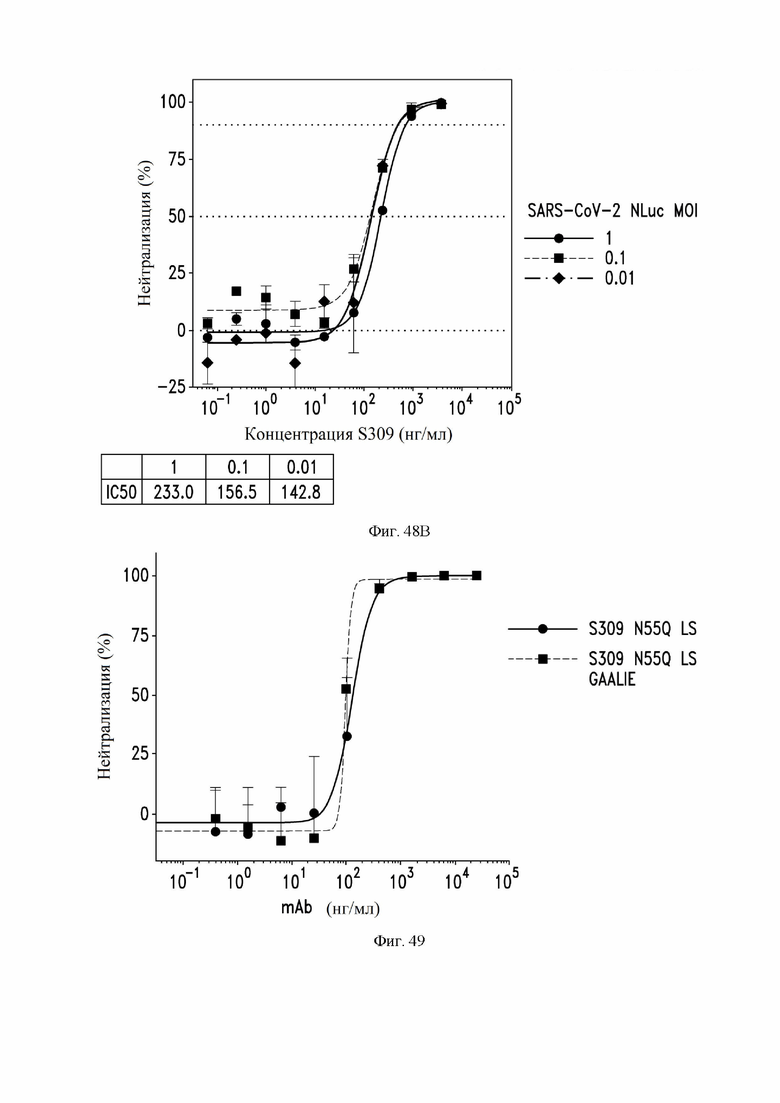
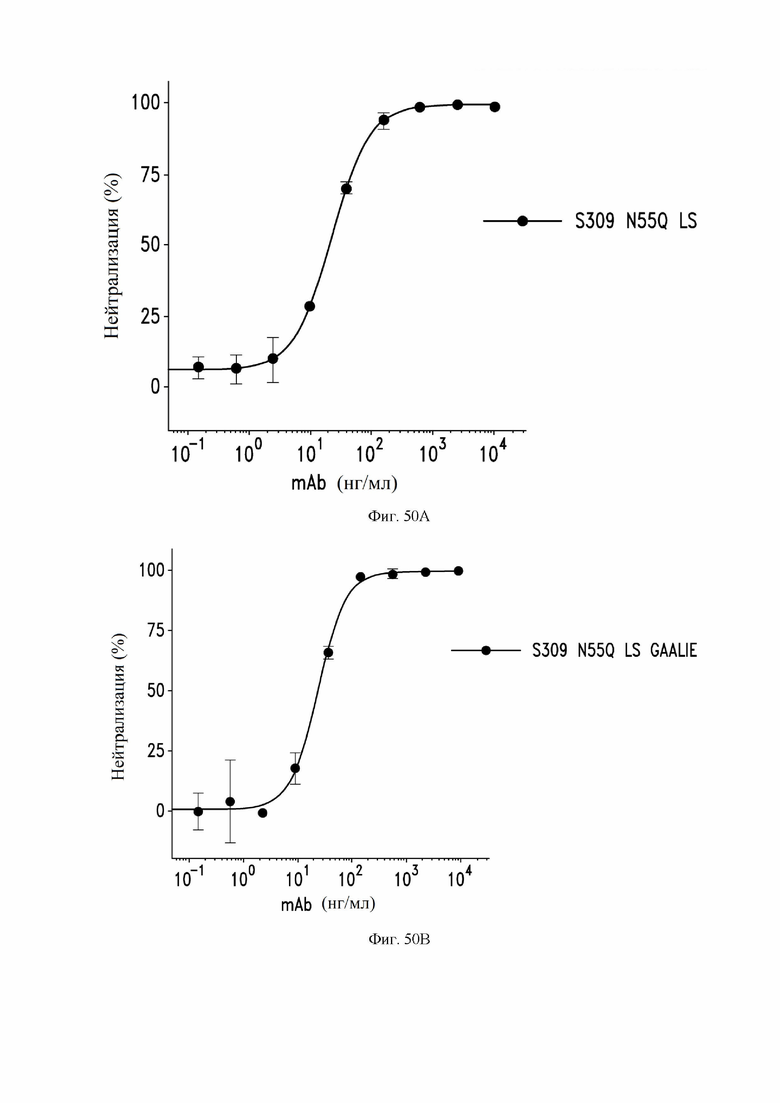
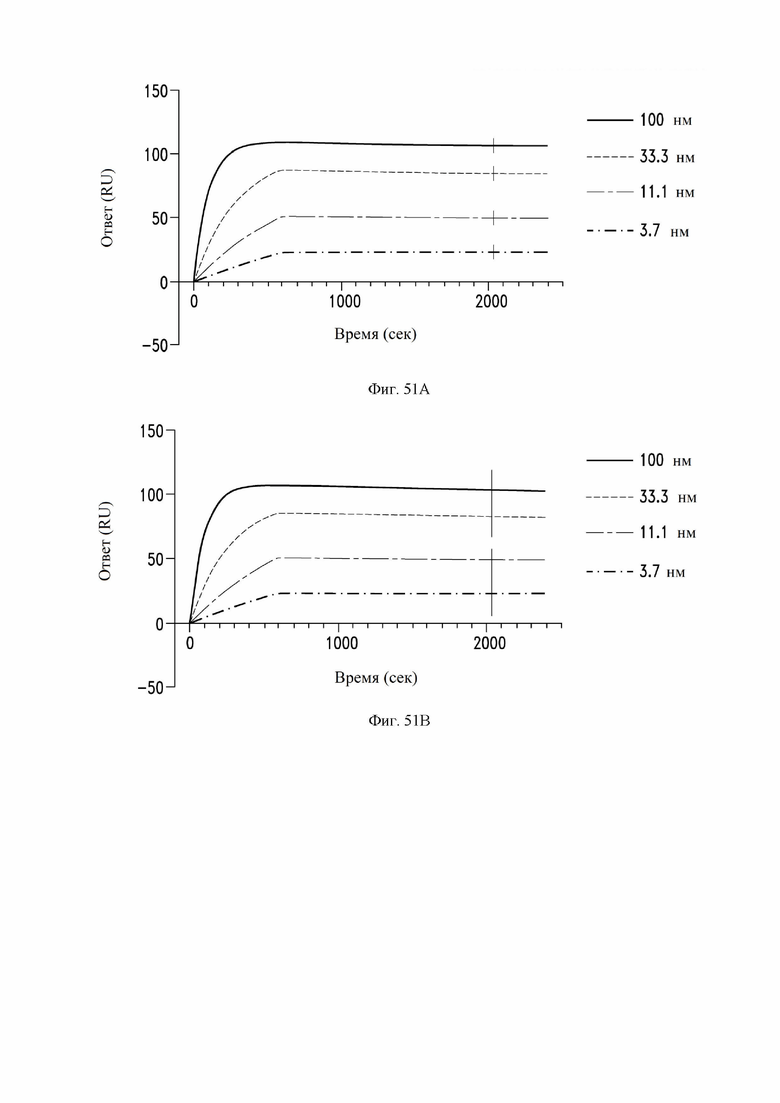
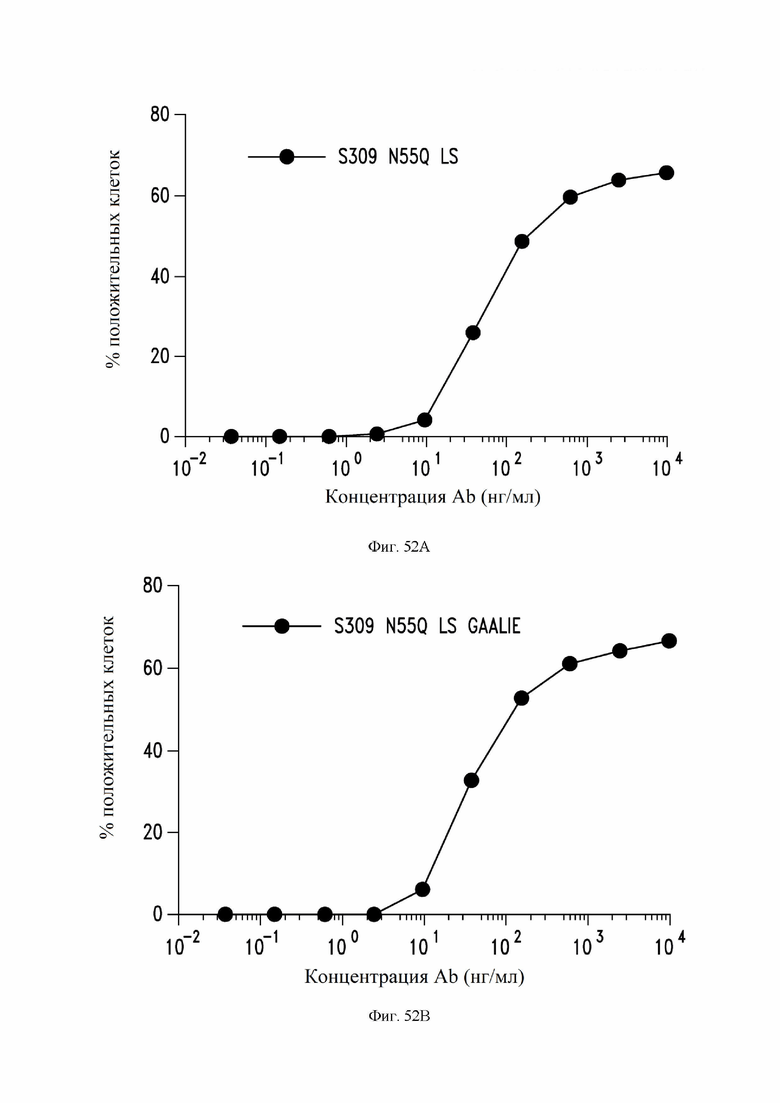
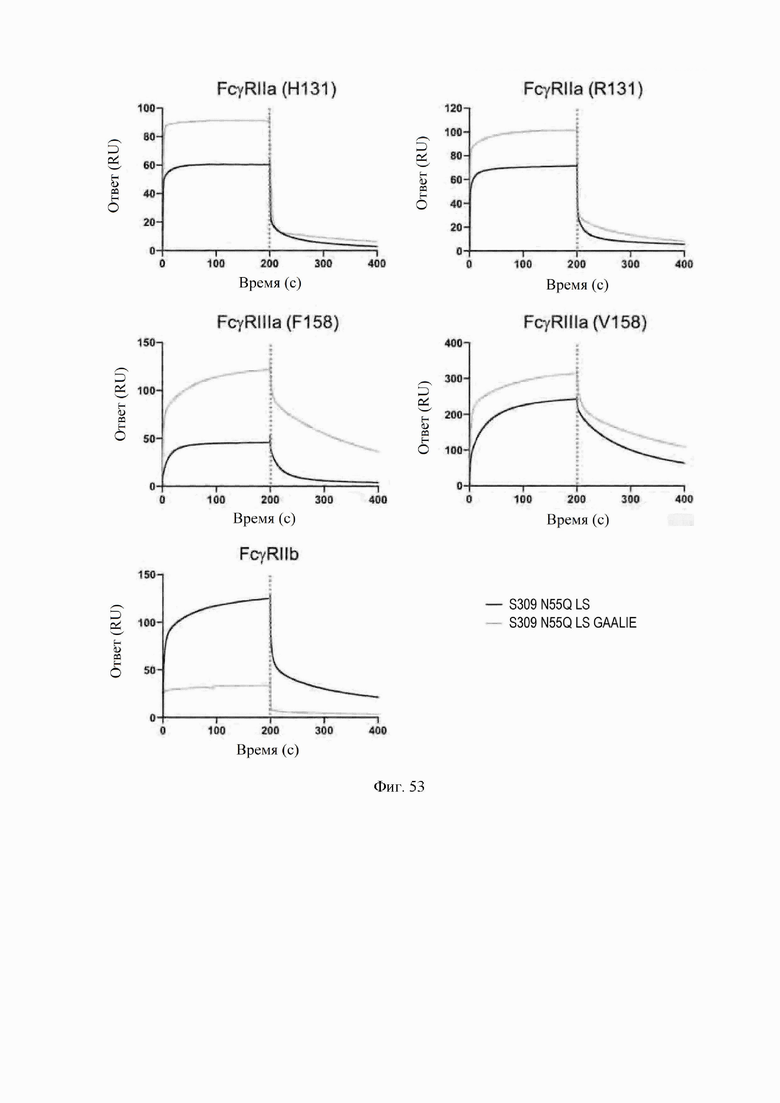
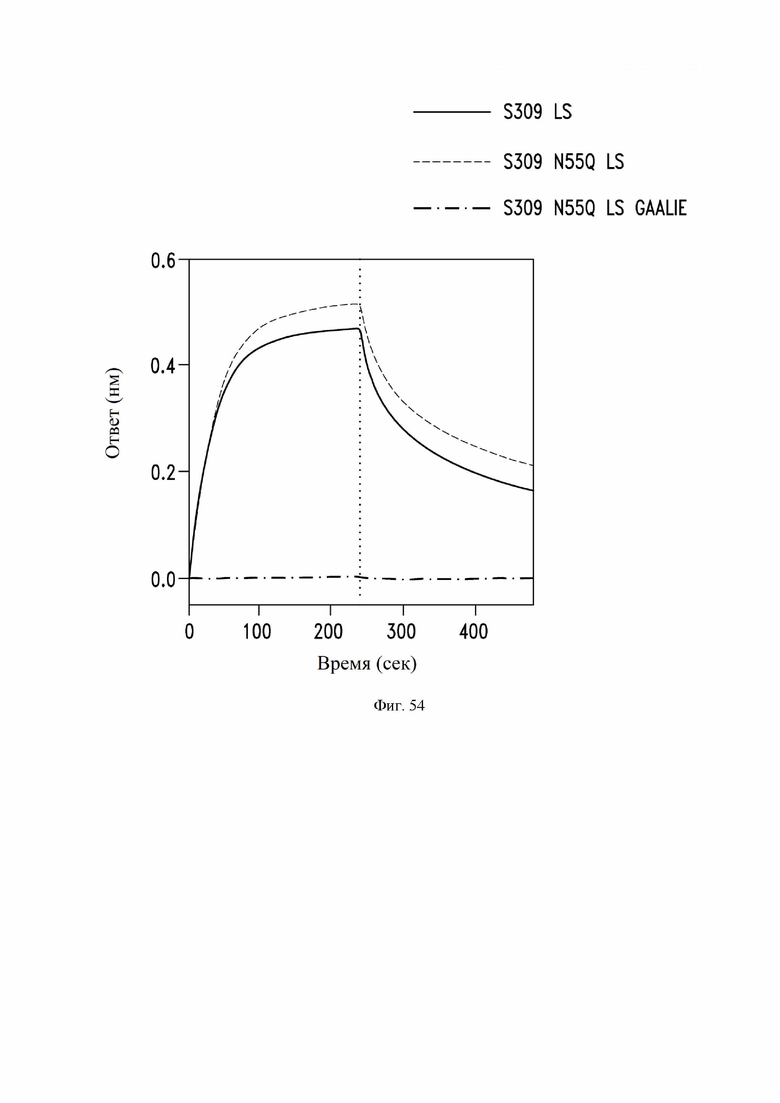
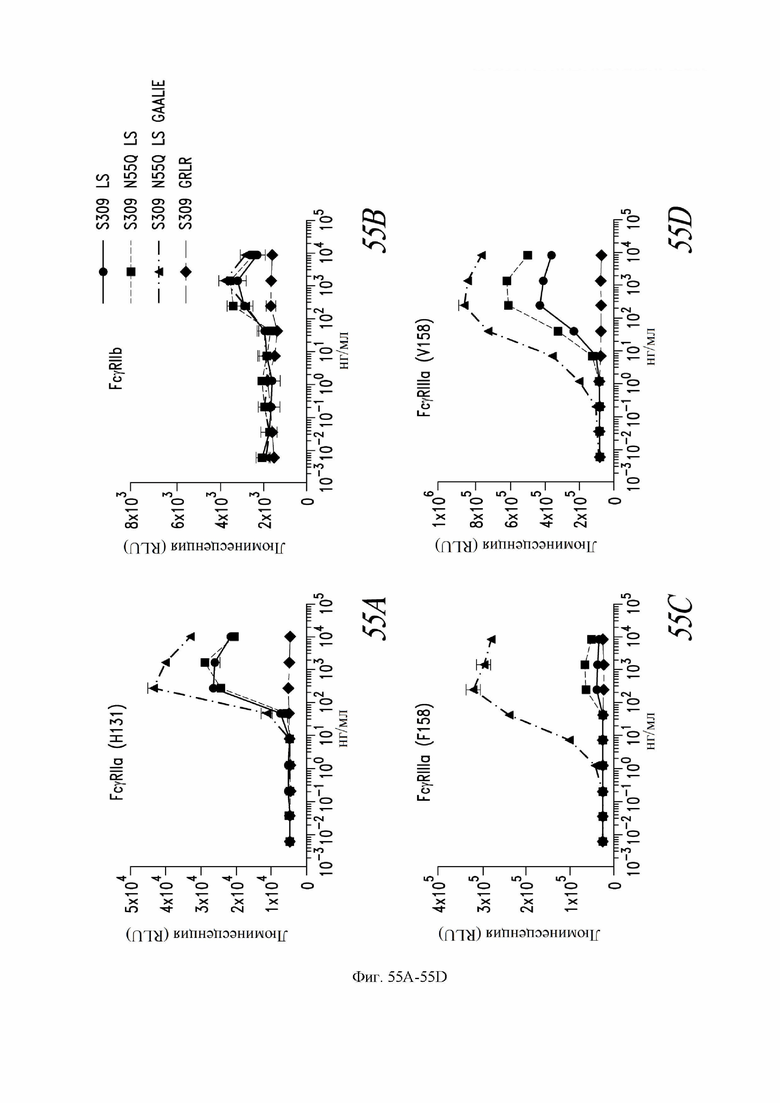
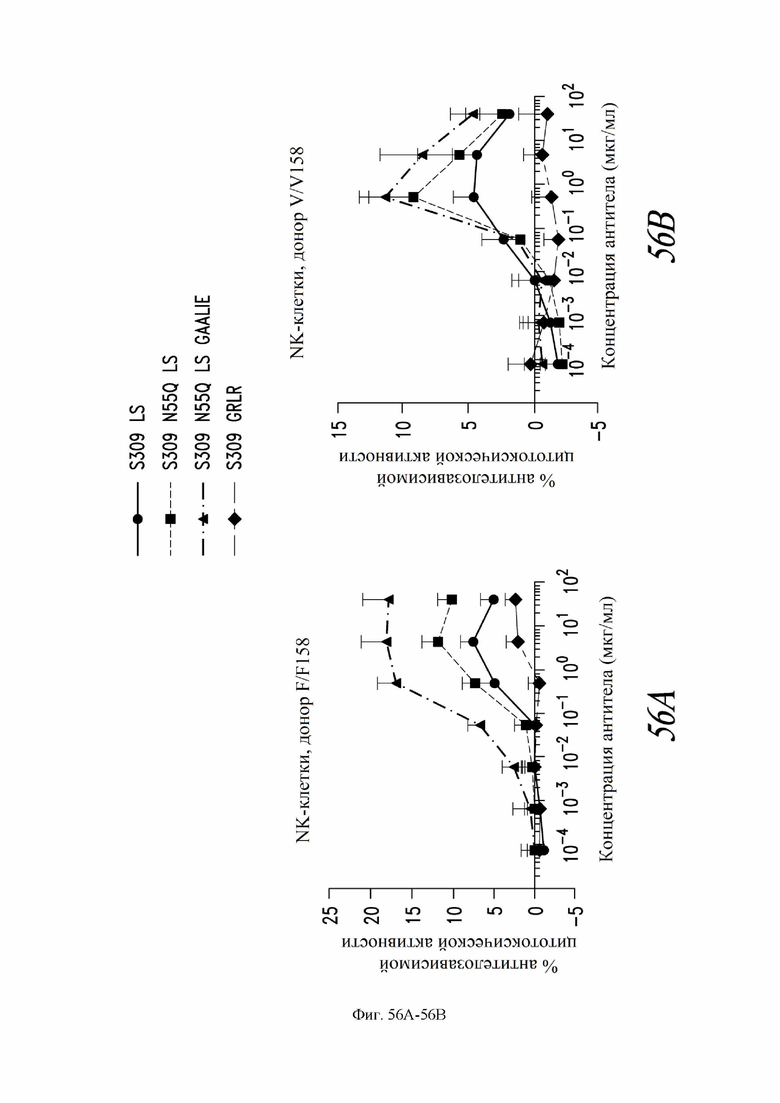
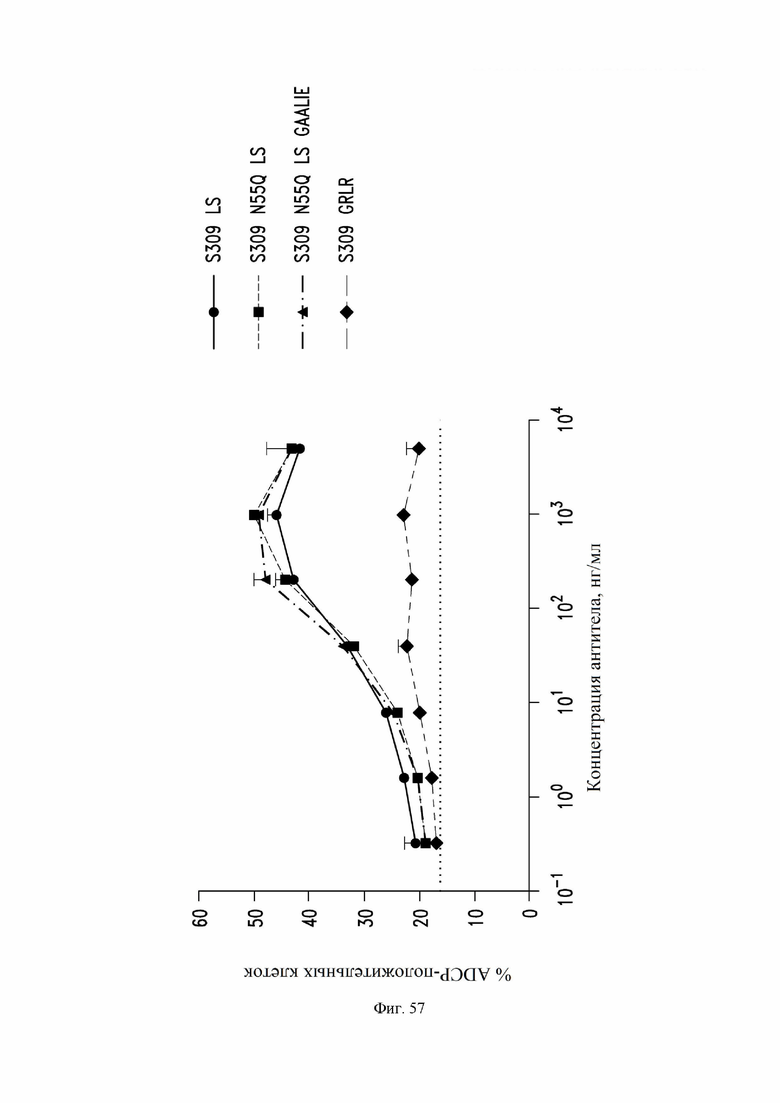
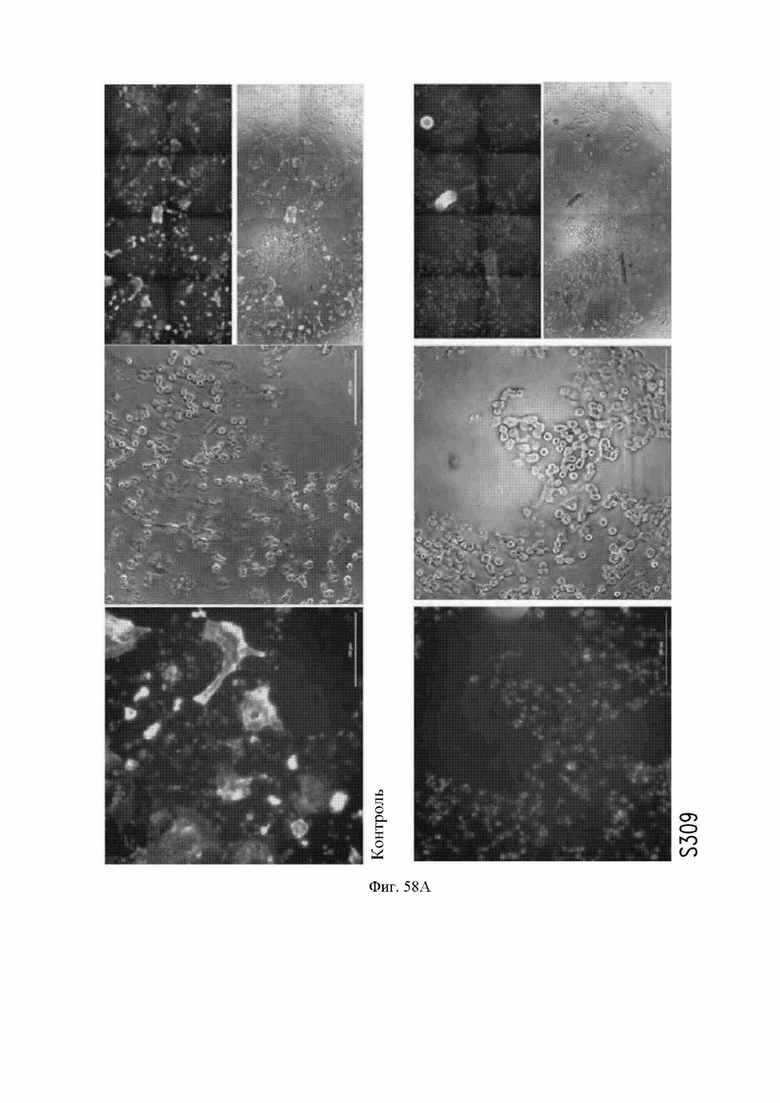
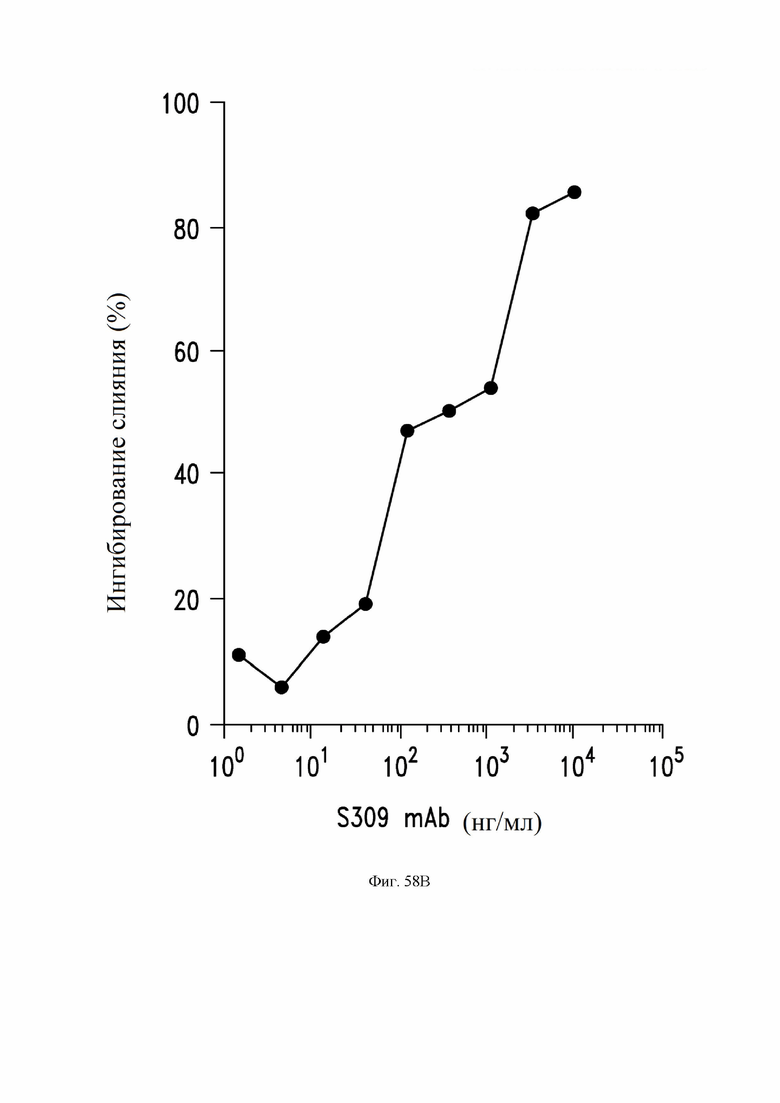
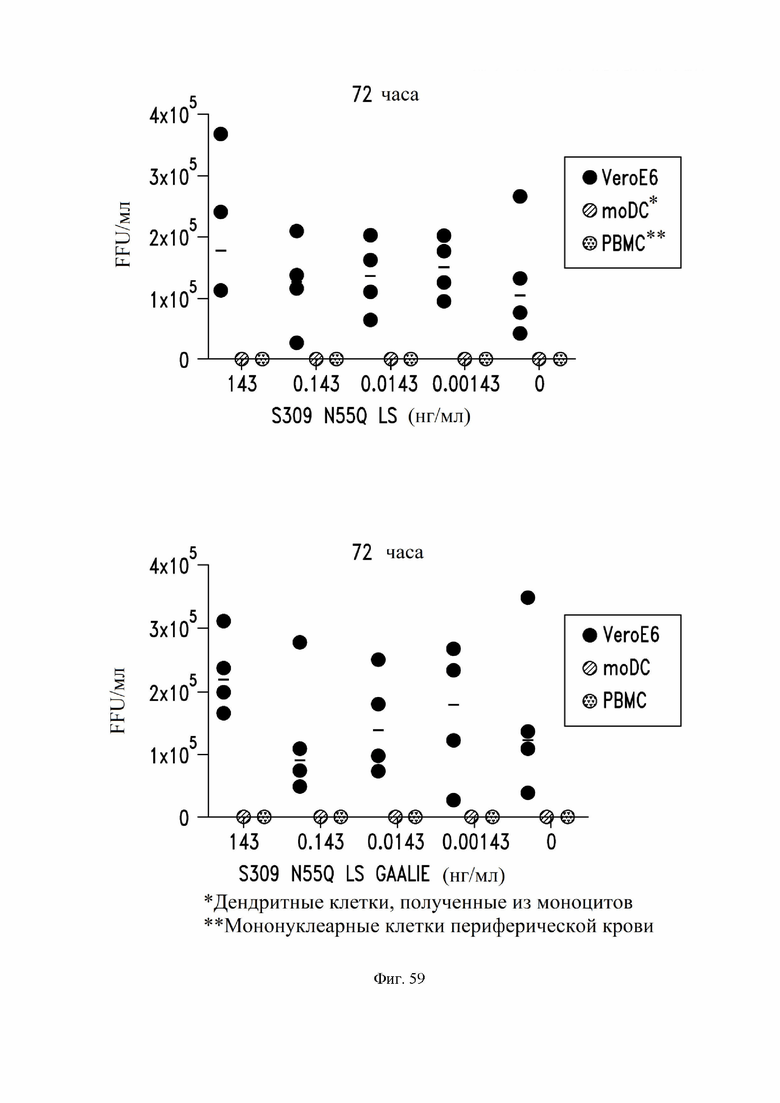
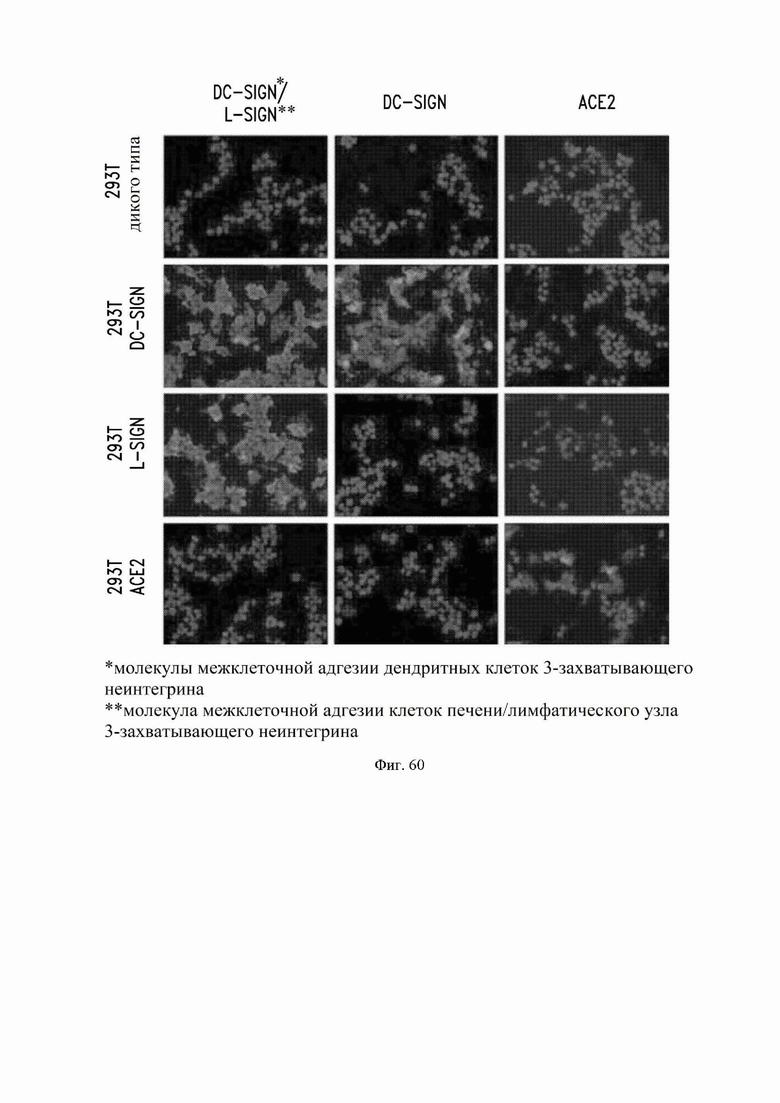
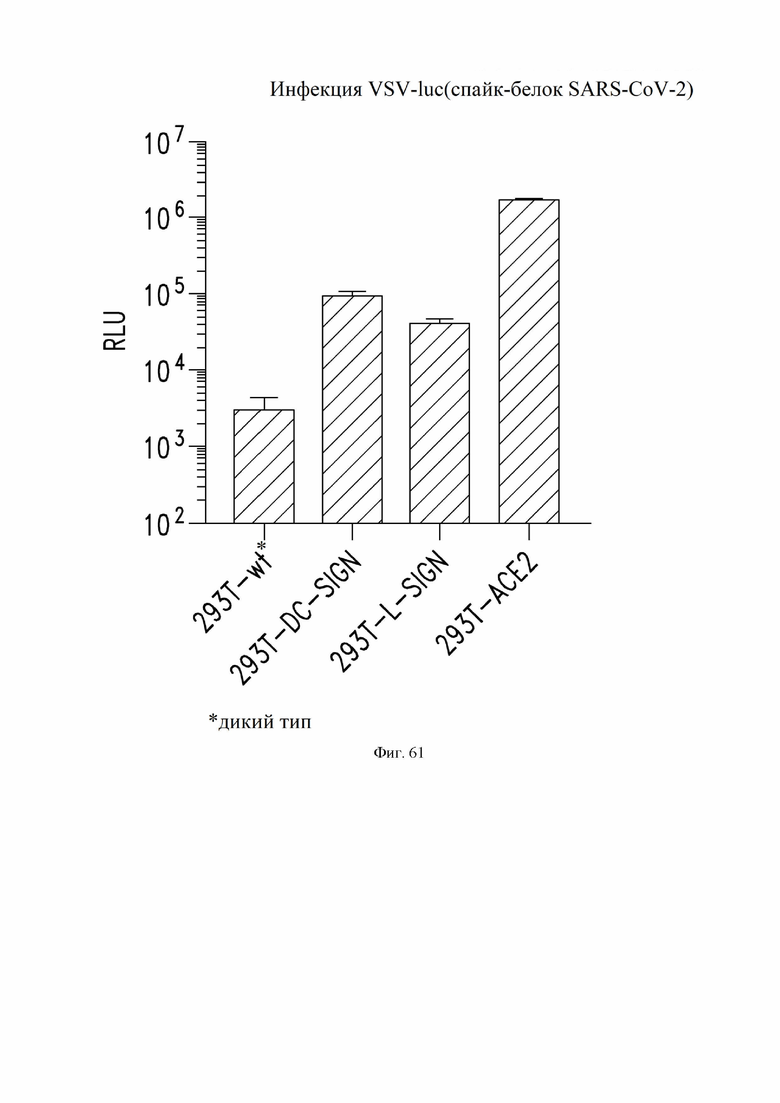
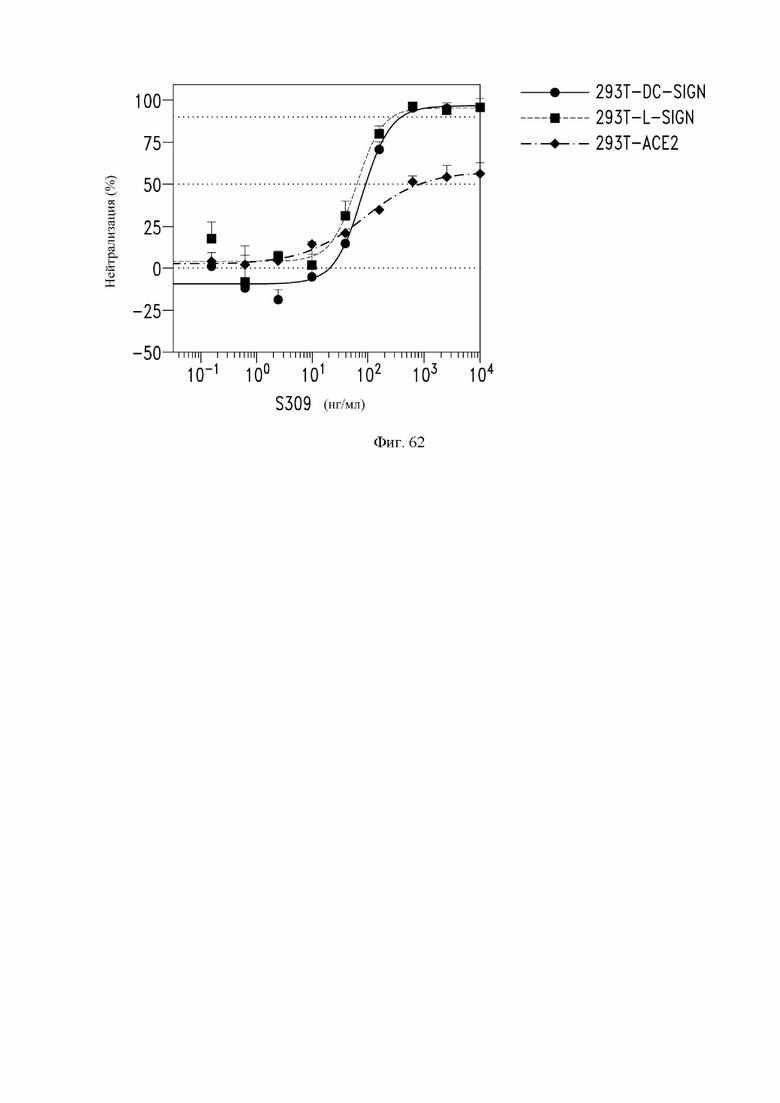
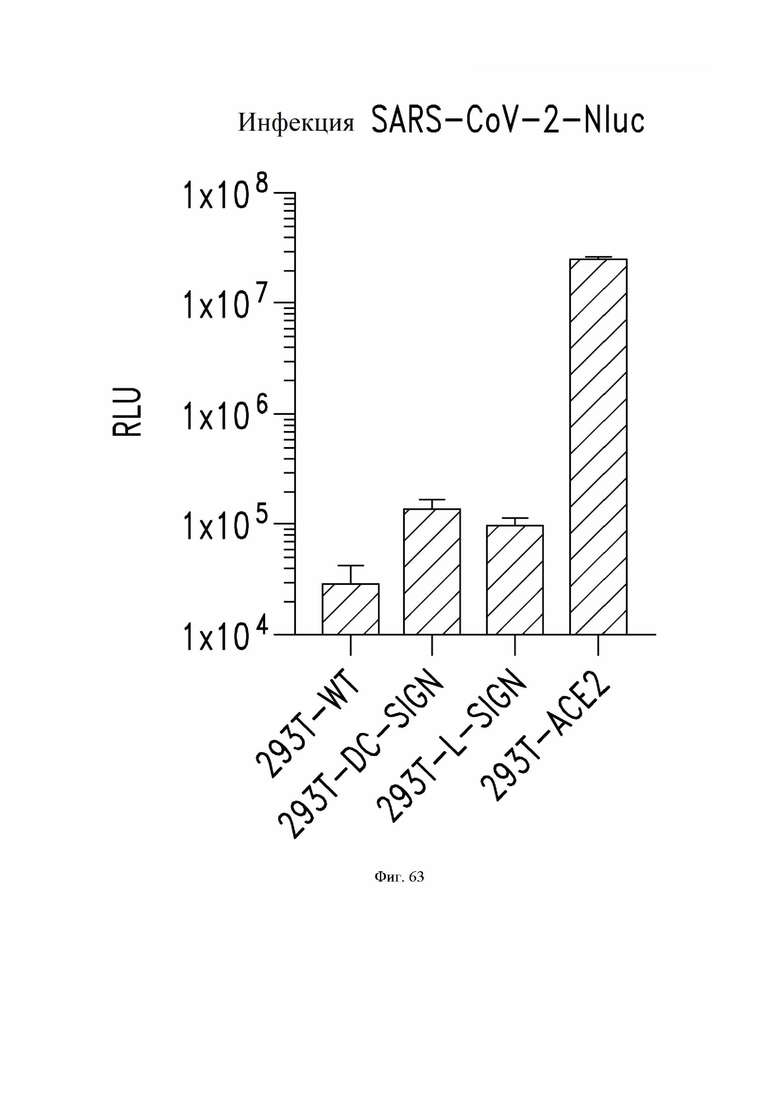
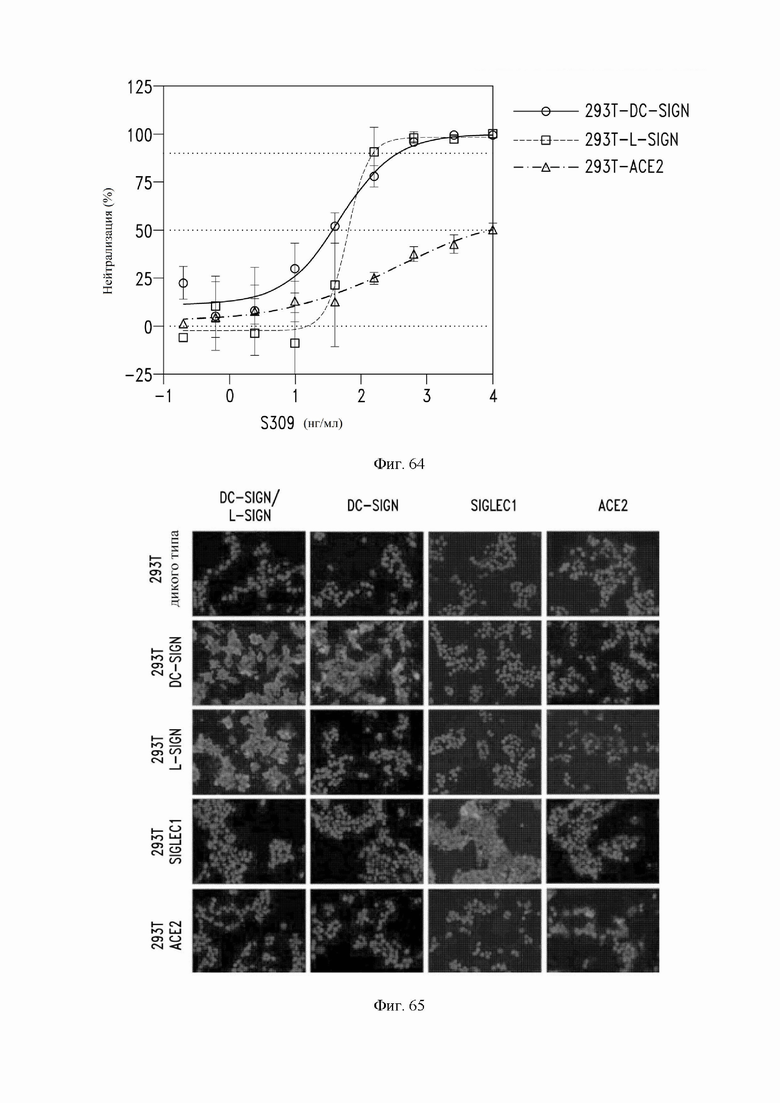
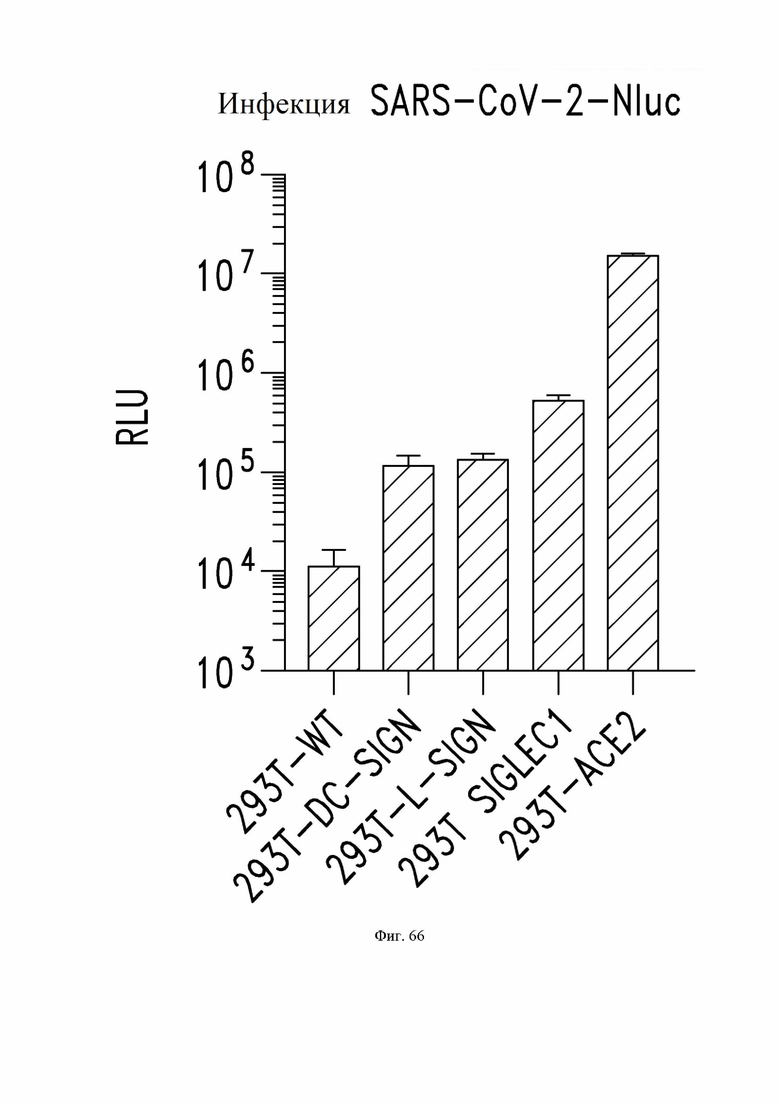
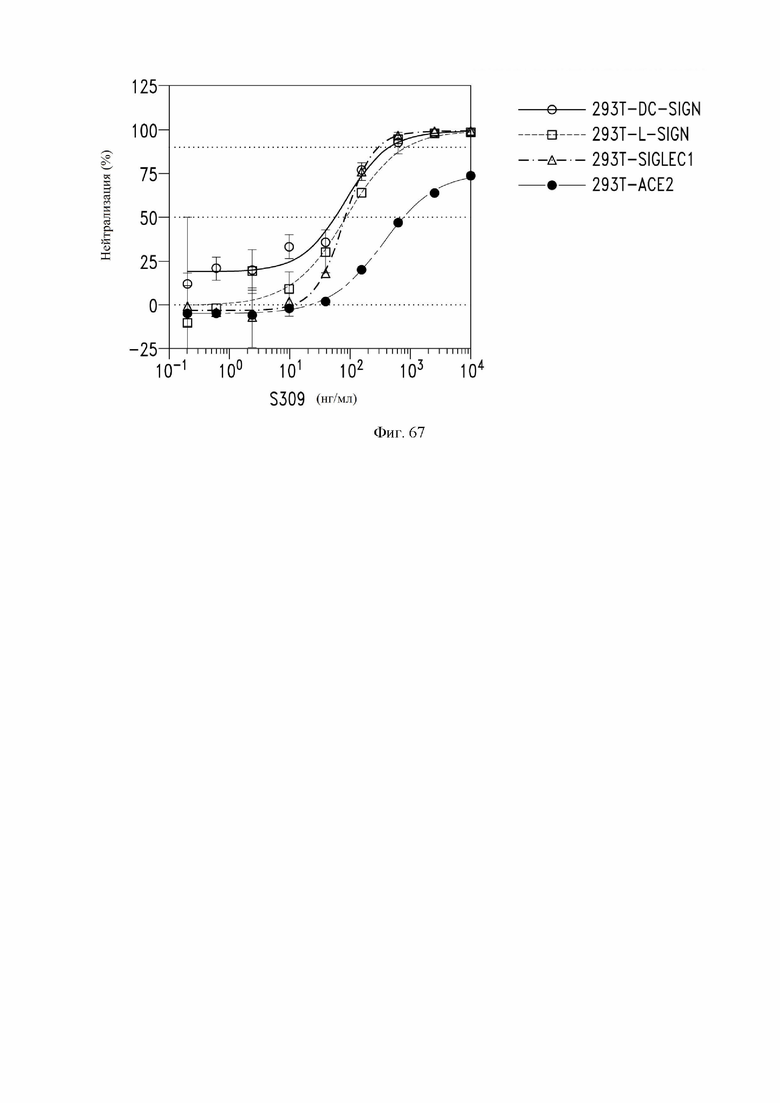

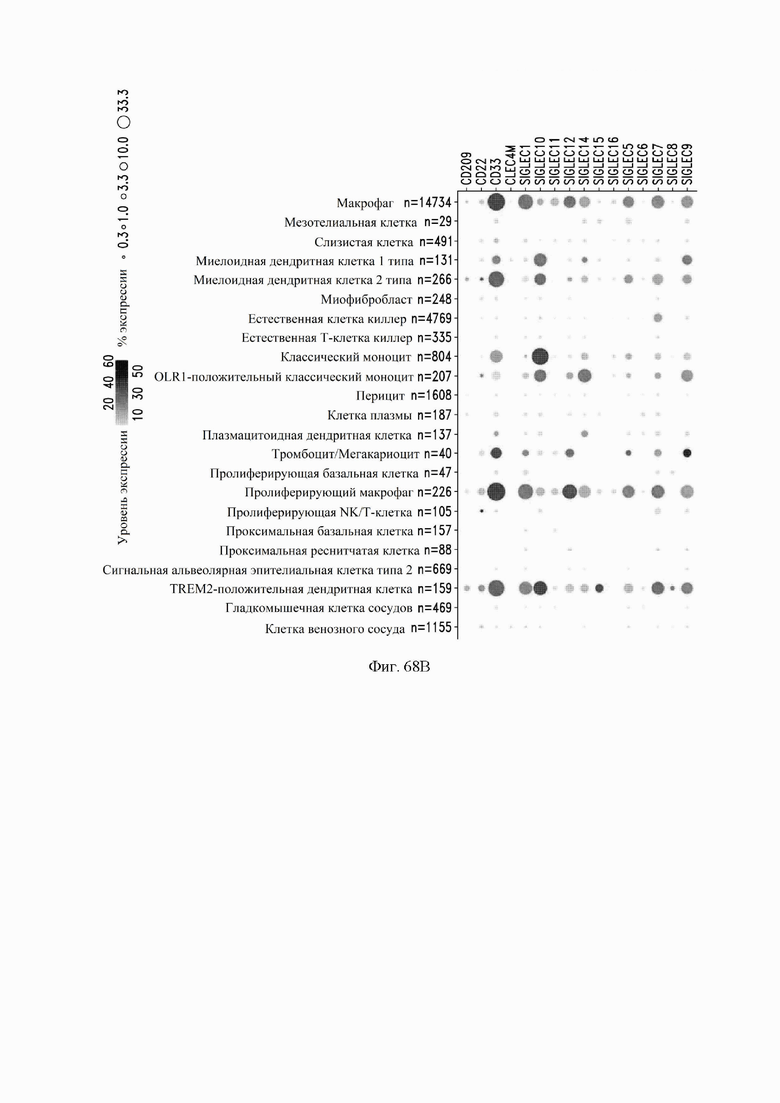
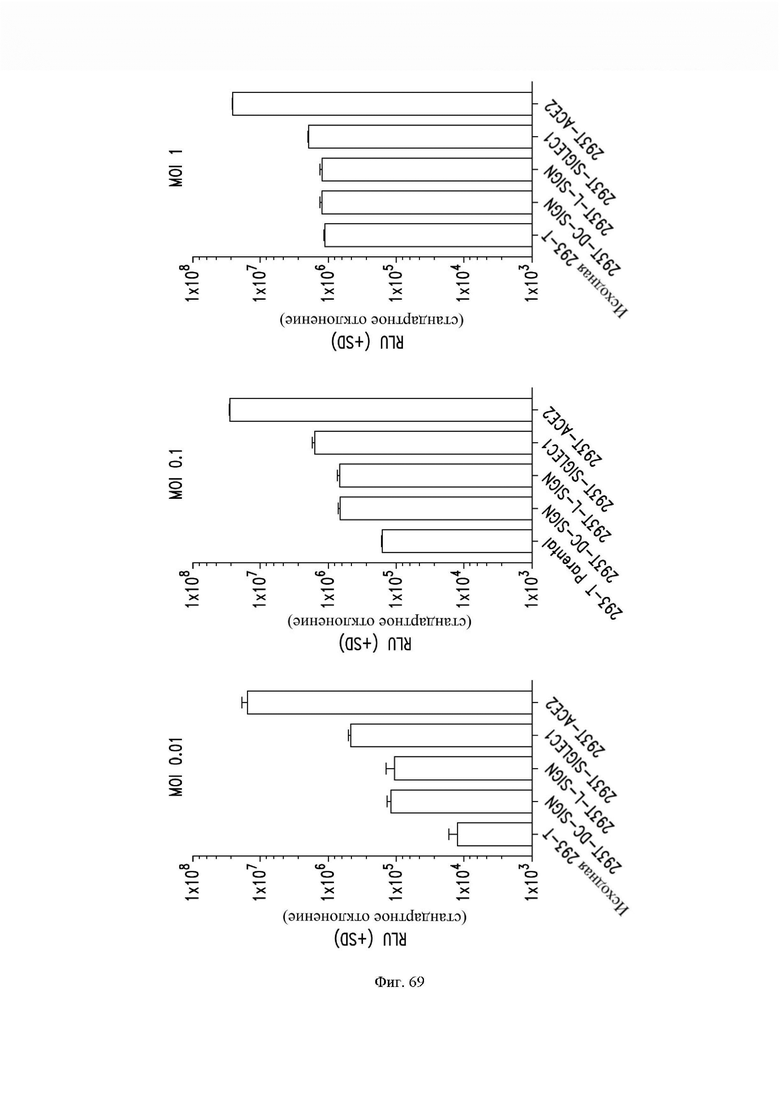

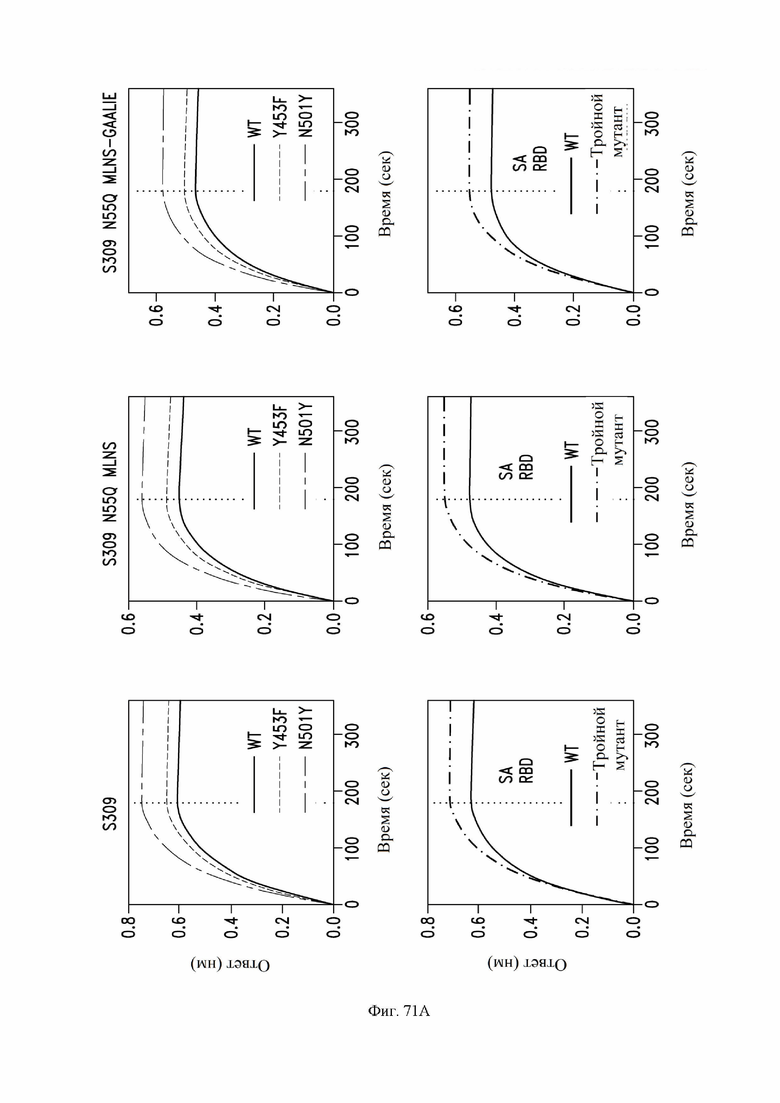
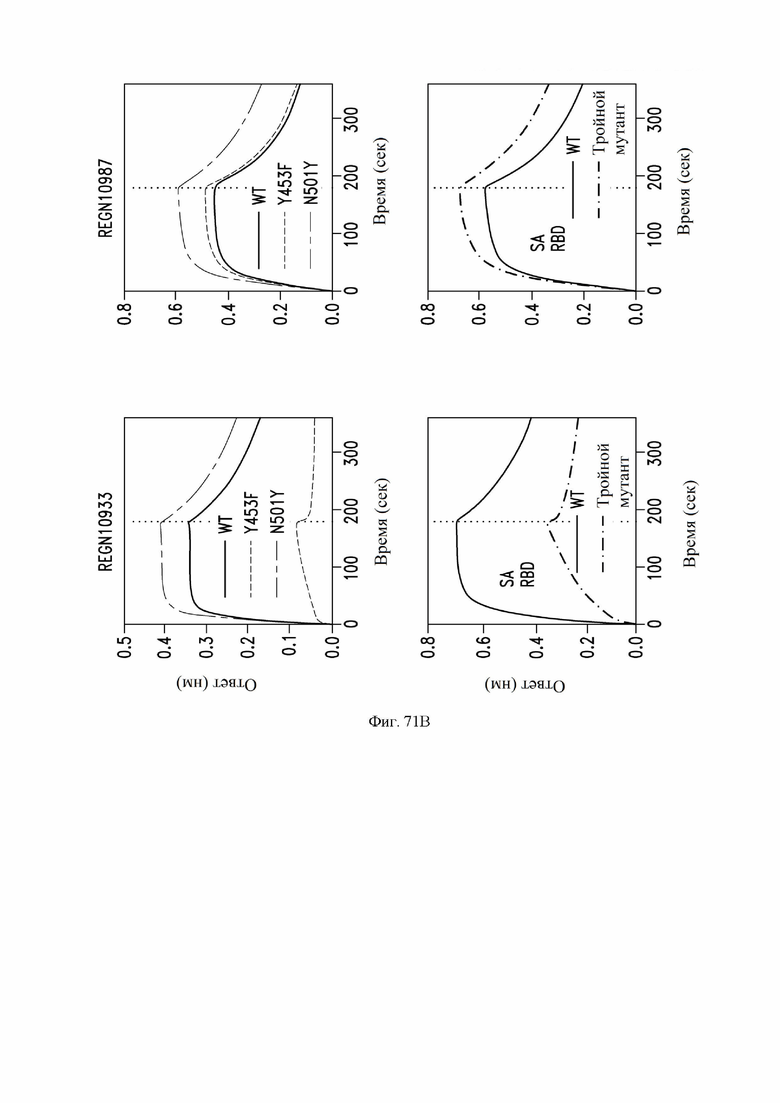
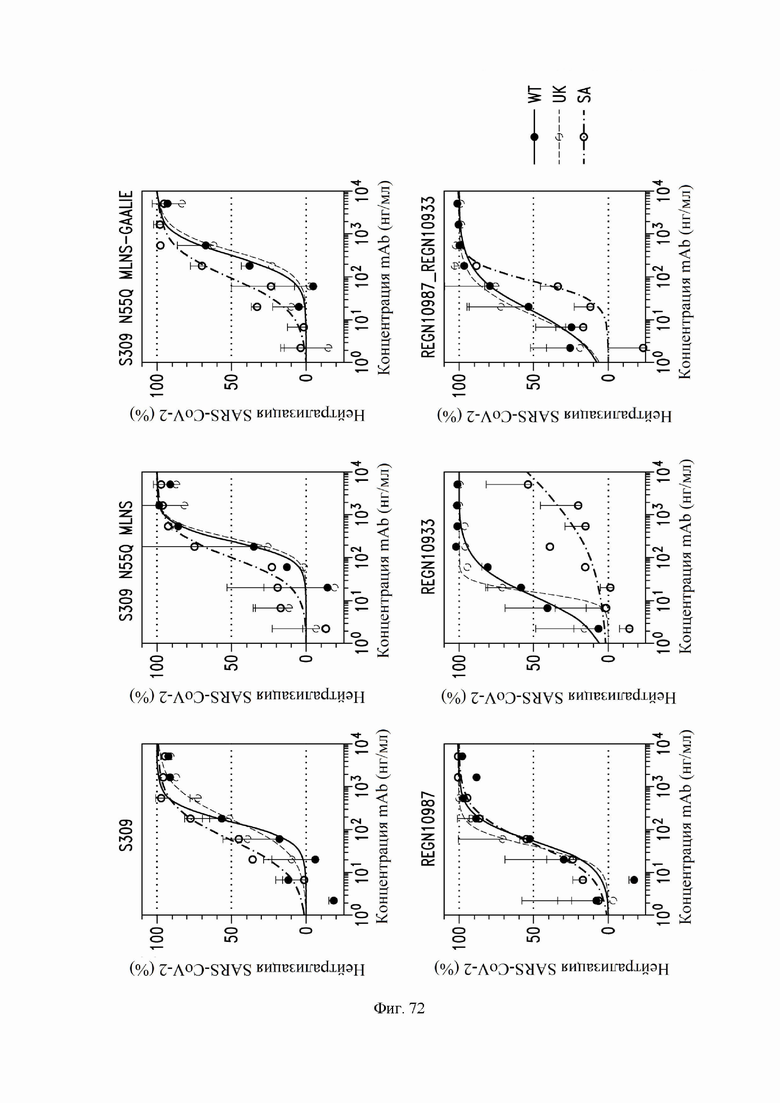
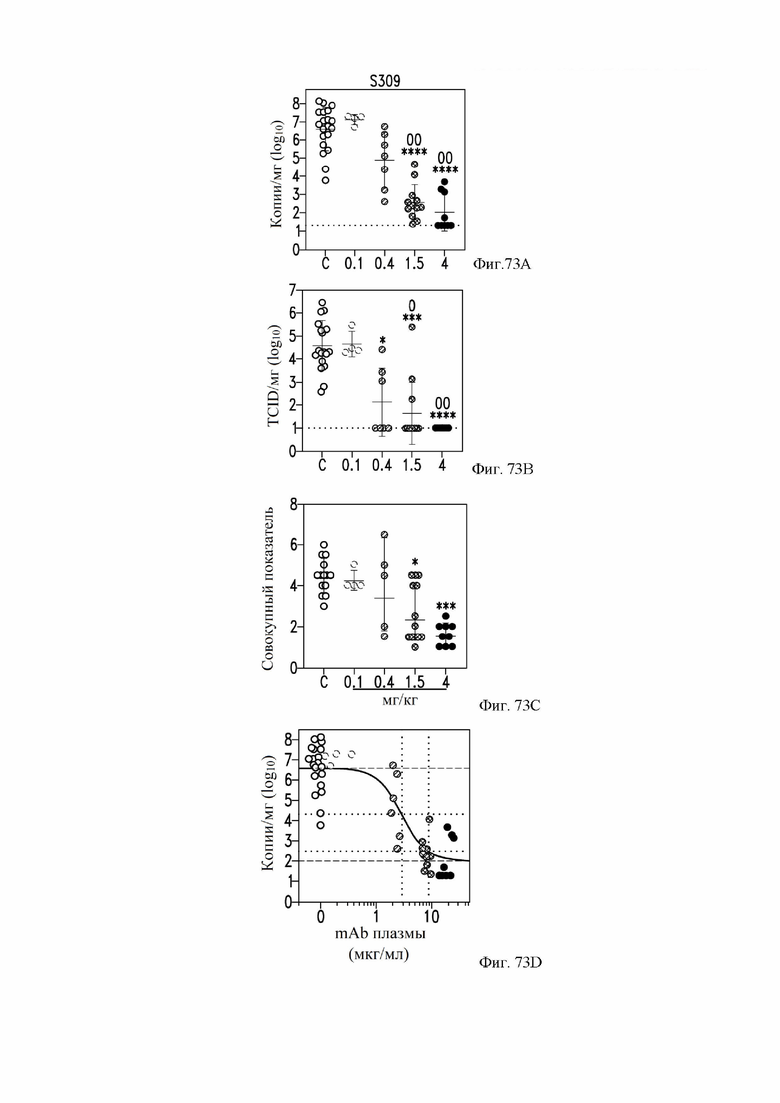
Настоящее изобретение относится к медицинской биотехнологии. Предложены антитела и их антигенсвязывающие фрагменты, которые могут связываться с антигеном SARS-CoV-2 (коронавирус-2 тяжелого острого респираторного синдрома) и в некоторых вариантах осуществления способны нейтрализовать инфекцию SARS-CoV-2. Также предложены полинуклеотиды, кодирующие антитело или антигенсвязывающий фрагмент, рекомбинантный вектор экспрессии для получения антитела, клетки-хозяева, содержащие полинуклеотид, фармацевтические композиции для лечения инфекции SARS-CoV-2, набор для доставки аэрозольной композиции и способы применения описанных представленых антител, антигенсвязывающих фрагментов и композиций в получении лекарственного средства для лечения инфекции SARS-CoV-2 у субъекта или диагностики инфекции SARS-CoV-2. Предложены также способ in vitro получения антитела и способ лечения инфекции SARS-CoV-2 у субъекта. Изобретение позволяет предотвращать или лечить инфекции SARS-CoV-2 или проводить диагностику инфекции SARS-CoV-2. 19 н. и 43 з.п. ф-лы, 148 ил., 13 табл., 39 пр.
1. Выделенное антитело или его антигенсвязывающий фрагмент, содержащие вариабельный домен тяжелой цепи (VH), содержащий аминокислотную последовательность области, определяющей комплементарность (CDR) H1, представленную в SEQ ID NO: 106, аминокислотную последовательность CDRH2, представленную в SEQ ID NO: 121, и аминокислотную последовательность CDRH3, представленную в SEQ ID NO: 108, и вариабельный домен легкой цепи (VL), содержащий аминокислотную последовательность CDRL1, представленную в SEQ ID NO: 169, аминокислотную последовательность CDRL2, представленную в SEQ ID NO: 170, и аминокислотную последовательность CDRL3, представленную в SEQ ID NO: 171, где антитело или антигенсвязывающий фрагмент способны связываться с поверхностным гликопротеином (S) (S - спайк-гликопротеин) SARS-CoV-2 (коронавирус-2 тяжелого острого респираторного синдрома).
2. Выделенное антитело или антигенсвязывающий фрагмент по п. 1, способные связываться с поверхностным гликопротеином (S) SARS-CoV-2, экспрессируемым на клеточной поверхности клетки-хозяина, на вирионе SARS-CoV-2 или на обоих.
3. Выделенное антитело или антигенсвязывающий фрагмент по п. 1 или 2, которые способны связываться с поверхностным гликопротеином (S): SARS-CoV-2 Wuhan-Hu-1, SEQ ID NO: 165.
4. Выделенное антитело или антигенсвязывающий фрагмент по любому из пп. 1-3, где:
(i) VH содержит или состоит из аминокислотной последовательности, имеющей по меньшей мере 85% идентичности с аминокислотной последовательностью, представленной в SEQ ID NO: 113; и/или
(ii) VL содержит или состоит из аминокислотной последовательности, имеющей по меньшей мере 85% идентичности с аминокислотной последовательностью, представленной в SEQ ID NO: 168.
5. Выделенное антитело или антигенсвязывающий фрагмент по п. 4, где:
(i) VH содержит или состоит из аминокислотной последовательности, имеющей по меньшей мере 90% идентичности с аминокислотной последовательностью, представленной в SEQ ID NO: 113; и/или
(ii) VL содержит или состоит из аминокислотной последовательности, имеющей по меньшей мере 90% идентичности с аминокислотной последовательностью, представленной в SEQ ID NO: 168.
6. Выделенное антитело или антигенсвязывающий фрагмент по п. 4, где:
(i) VH содержит или состоит из аминокислотной последовательности, имеющей по меньшей мере 95% идентичности с аминокислотной последовательностью, представленной в SEQ ID NO: 113; и/или
(ii) VL содержит или состоит из аминокислотной последовательности, имеющей по меньшей мере 95% идентичности с аминокислотной последовательностью, представленной в SEQ ID NO: 168.
7. Выделенное антитело или антигенсвязывающий фрагмент по п. 4, где:
(i) VH содержит или состоит из аминокислотной последовательности, имеющей по меньшей мере 99% идентичности с аминокислотной последовательностью, представленной в SEQ ID NO: 113; и/или
(ii) VL содержит или состоит из аминокислотной последовательности, имеющей по меньшей мере 99% идентичности с аминокислотной последовательностью, представленной в SEQ ID NO: 168.
8. Выделенное антитело или его антигенсвязывающий фрагмент, содержащие вариабельный домен тяжелой цепи (VH) и вариабельный домен легкой цепи (VL), где VH содержит или состоит из аминокислотной последовательности, представленной в SEQ ID NO: 113, и VL содержит или состоит из аминокислотной последовательности, представленной в SEQ ID NO: 168, где антитело или антигенсвязывающий фрагмент способны связываться с поверхностным гликопротеином (S) SARS-CoV-2.
9. Выделенное антитело или его антигенсвязывающий фрагмент, содержащие вариабельный домен тяжелой цепи (VH), содержащий аминокислотную последовательность области, определяющей комплементарность (CDR) H1, представленную в SEQ ID NO: 106, аминокислотную последовательность CDRH2, представленную в SEQ ID NO: 107, и аминокислотную последовательность CDRH3, представленную в SEQ ID NO: 108, и вариабельный домен легкой цепи (VL), содержащий аминокислотную последовательность CDRL1, представленную в SEQ ID NO: 169, аминокислотную последовательность CDRL2, представленную в SEQ ID NO: 170, и аминокислотную последовательность CDRL3, представленную в SEQ ID NO: 171, где антитело или антигенсвязывающий фрагмент способны связываться с поверхностным гликопротеином (S) SARS-CoV-2.
10. Выделенное антитело или антигенсвязывающий фрагмент по п. 9, способные связываться с поверхностным гликопротеином (S) SARS-CoV-2, экспрессируемым на клеточной поверхности клетки-хозяина, на вирионе SARS-CoV-2 или на обоих.
11. Выделенное антитело или антигенсвязывающий фрагмент по п. 9 или 10, которые способны связываться с поверхностным гликопротеином (S): SARS-CoV-2 Wuhan-Hu-1, SEQ ID NO: 165.
12. Выделенное антитело или антигенсвязывающий фрагмент по любому из пп. 9-11, где:
(i) VH содержит или состоит из аминокислотной последовательности, имеющей по меньшей мере 85% идентичности с аминокислотной последовательностью, представленной в SEQ ID NO: 105; и/или
(ii) VL содержит или состоит из аминокислотной последовательности, имеющей по меньшей мере 85% идентичности с аминокислотной последовательностью, представленной в SEQ ID NO: 168.
13. Выделенное антитело или антигенсвязывающий фрагмент по п. 12, где:
(i) VH содержит или состоит из аминокислотной последовательности, имеющей по меньшей мере 90% идентичности с аминокислотной последовательностью, представленной в SEQ ID NO: 105; и/или
(ii) VL содержит или состоит из аминокислотной последовательности, имеющей по меньшей мере 90% идентичности с аминокислотной последовательностью, представленной в SEQ ID NO: 168.
14. Выделенное антитело или антигенсвязывающий фрагмент по п. 12, где:
(i) VH содержит или состоит из аминокислотной последовательности, имеющей по меньшей мере 95% идентичности с аминокислотной последовательностью, представленной в SEQ ID NO: 105; и/или
(ii) VL содержит или состоит из аминокислотной последовательности, имеющей по меньшей мере 95% идентичности с аминокислотной последовательностью, представленной в SEQ ID NO: 168.
15. Выделенное антитело или антигенсвязывающий фрагмент по п. 12, где:
(i) VH содержит или состоит из аминокислотной последовательности, имеющей по меньшей мере 99% идентичности с аминокислотной последовательностью, представленной в SEQ ID NO: 105; и/или
(ii) VL содержит или состоит из аминокислотной последовательности, имеющей по меньшей мере 99% идентичности с аминокислотной последовательностью, представленной в SEQ ID NO: 168.
16. Выделенное антитело или его антигенсвязывающий фрагмент, содержащие вариабельный домен тяжелой цепи (VH) и вариабельный домен легкой цепи (VL), где VH содержит или состоит из аминокислотной последовательности, представленной в SEQ ID NO: 105, и VL содержит или состоит из аминокислотной последовательности, представленной в SEQ ID NO: 168, где антитело или антигенсвязывающий фрагмент способны связываться с поверхностным гликопротеином (S) SARS-CoV-2.
17. Выделенное антитело или антигенсвязывающий фрагмент по любому из пп. 1-16, которые способны нейтрализовать инфекцию SARS-CoV-2:
(i) на модели инфекции in vitro;
(ii) на животной модели инфекции in vivo;
(iii) у человека; или
(iv) любой комбинации (i)-(iii).
18. Выделенное антитело или антигенсвязывающий фрагмент по любому из пп. 1-17, где антитело или антигенсвязывающий фрагмент включает антитело человека, моноклональное антитело, очищенное антитело, одноцепочечное антитело, Fab, Fab', F(ab')2, Fv, scFv или scFab.
19. Выделенное антитело или антигенсвязывающий фрагмент по любому из пп. 1-18, где антитело или антигенсвязывающий фрагмент дополнительно содержит полипептид Fc или его фрагмент.
20. Выделенное антитело или антигенсвязывающий фрагмент по п. 19, которое представляет собой изотип IgG, IgA, IgM, IgE или IgD.
21. Выделенное антитело или антигенсвязывающий фрагмент по п. 20, которое представляет собой изотип IgG1.
22. Выделенное антитело или антигенсвязывающий фрагмент по любому из пп. 19-21, где полипептид Fc или его фрагмент содержит:
(i) мутацию, усиливающую связывание с FcRn по сравнению с эталонным полипептидом Fc, который не содержит мутацию; и/или
(ii) мутацию, усиливающую связывание с FcγR по сравнению с эталонным полипептидом Fc, который не содержит мутацию.
23. Выделенное антитело или антигенсвязывающий фрагмент по п. 22, где мутация, усиливающая связывание с FcRn, включает:
(i) M428L/N434S;
(ii) M252Y/S254T/T256E;
(iii) T250Q/M428L;
(iv) P257I/Q311I;
(v) P257I/N434H;
(vi) D376V/N434H;
(vii) T307A/E380A/N434A или
(viii) любую комбинацию (i)-(vii).
24. Выделенное антитело или антигенсвязывающий фрагмент по п. 23, где мутация, усиливающая связывание с FcRn, включает M428L/N434S.
25. Выделенное антитело или антигенсвязывающий фрагмент по любому из пп. 22-24, где мутация, усиливающая связывание с FcγR, включает S239D, I332E, A330L, G236A или любую их комбинацию.
26. Выделенное антитело или антигенсвязывающий фрагмент по п. 25, где мутация, усиливающая связывание с FcγR, включает:
(i) S239D/I332E;
(ii) S239D/A330L/I332E;
(iii) G236A/S239D/I332E или
(iv) G236A/A330L/I332E.
27. Выделенное антитело или антигенсвязывающий фрагмент по любому из пп. 1-26, дополнительно содержащие CH1-CH3, имеющую 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентичности с, или содержащую, или состоящую из аминокислотной последовательности, представленной в SEQ ID NO: 173 или 175.
28. Выделенное антитело или антигенсвязывающий фрагмент по любому из пп. 1-27, дополнительно содержащие CL, имеющую 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентичности с, или содержащую, или состоящую из аминокислотной последовательности, представленной в SEQ ID NO: 174.
29. Выделенное антитело, содержащее:
(i) тяжелую цепь, содержащую (i)(1) VH, содержащий или состоящий из аминокислотной последовательности, представленной в SEQ ID NO: 113, и (i)(2) CH1-CH3, содержащую или состоящую из аминокислотной последовательности, представленной в SEQ ID NO: 173; и
(ii) легкую цепь, содержащую (ii)(1) VL, содержащий или состоящий из аминокислотной последовательности, представленной в SEQ ID NO: 168, и (ii)(2) CL, содержащую или состоящую из аминокислотной последовательности, представленной в SEQ ID NO: 174, где антитело способно связываться с поверхностным гликопротеином (S) SARS-CoV-2.
30. Выделенное антитело, содержащее:
(i) тяжелую цепь, содержащую (i)(1) VH, содержащий или состоящий из аминокислотной последовательности, представленной в SEQ ID NO: 113, и (i)(2) CH1-CH3, содержащую или состоящую из аминокислотной последовательности, представленной в SEQ ID NO: 175; и
(ii) легкую цепь, содержащую (ii)(1) VL, содержащий или состоящий из аминокислотной последовательности, представленной в SEQ ID NO: 168, и (ii)(2) CL, содержащую или состоящую из аминокислотной последовательности, представленной в SEQ ID NO: 174, где антитело способно связываться с поверхностным гликопротеином (S) SARS-CoV-2.
31. Выделенное антитело, содержащее:
(i) тяжелую цепь, содержащую (i)(1) VH, содержащий или состоящий из аминокислотной последовательности, представленной в SEQ ID NO: 105, и (i)(2) CH1-CH3, содержащую или состоящую из аминокислотной последовательности, представленной в SEQ ID NO: 175; и
(ii) легкую цепь, содержащую (ii)(1) VL, содержащий или состоящий из аминокислотной последовательности, представленной в SEQ ID NO: 168, и (ii)(2) CL, содержащую или состоящую из аминокислотной последовательности, представленной в SEQ ID NO: 174, где антитело способно связываться с поверхностным гликопротеином (S) SARS-CoV-2.
32. Выделенный полинуклеотид, кодирующий антитело по любому из пп. 1-31 или антигенсвязывающий фрагмент по любому из пп. 1-28.
33. Выделенный полинуклеотид по п. 32, где полинуклеотид содержит дезоксирибонуклеиновую кислоту (ДНК) или рибонуклеиновую кислоту (РНК), где РНК необязательно включает матричную РНК (мРНК).
34. Выделенный полинуклеотид по п. 33, где полинуклеотид содержит или состоит из мРНК.
35. Выделенный полинуклеотид по любому из пп. 32-34, который является кодон-оптимизированным для экспрессии в клетке-хозяине.
36. Выделенный полинуклеотид по п. 35, который явлется кодон-оптимизированным для экспрессии в клетке-хозяине млекопитающего.
37. Рекомбинантный вектор экспрессии для получения антитела по любому из пп. 1-31 или антигенсвязывающего фрагмента по любому из пп. 1-28, содержащий выделенный полинуклеотид по любому из пп. 32-36.
38. Клетка-хозяин для получения антитела по любому из пп. 1-31 или антигенсвязывающего фрагмента по любому из пп. 1-28, содержащая выделенный полинуклеотид по любому из пп. 32-36 и/или вектор по п. 37, где полинуклеотид является гетерологичным по отношению к клетке-хозяину.
39. В-клетка человека для получения антитела по любому из пп. 1-31 или антигенсвязывающего фрагмента по любому из пп. 1-28, содержащая выделенный полинуклеотид по любому из пп. 32-36, где полинуклеотид является гетерологичным по отношению к В-клетке человека, и/или где В-клетка человека является иммортализованной.
40. Фармацевтическая композиция для лечения инфекции SARS-CoV-2, содержащая:
(i) антитело по любому из пп. 1-31 или антигенсвязывающий фрагмент по любому из пп. 1-28;
(ii) выделенный полинуклеотид по любому из пп. 32-36;
(iii) рекомбинантный вектор по п. 37;
(iv) клетку-хозяина по п. 38 или
(v) В-клетку человека по п. 39
и фармацевтически приемлемое вспомогательное вещество, носитель или разбавитель.
41. Аэрозольная композиция для лечения инфекции SARS-CoV-2, содержащая композицию по п. 40.
42. Набор для доставки аэрозольной композиции по п. 41, включающий контейнер, содержащий аэрозольную композицию по п. 41 и одно или более из: активатора, клапана и субконтейнера.
43. Контейнер, содержащий:
(i) множество дозированных единиц аэрозольной композиции по п. 41, где композиция содержит антитело по любому из пп. 1-31 или антигенсвязывающий фрагмент по любому из пп. 1-28; или
(ii) композицию по п. 40, составленную для парентерального введения, где, необязательно, контейнер включает ампулу, одноразовый шприц или флакон с несколькими дозами.
44. Композиция по п. 40 или 41, содержащая выделенный полинуклеотид по любому из пп. 32-36, инкапсулированный в молекулу-носитель, где молекула-носитель необязательно включает липид, носитель, полученный из липида, такой как липосома, твердая липидная наночастица, маслянистая суспензия, субмикронная липидная эмульсия, липидный микропузырь, обратная липидная мицелла, кохлеарная липосома, липидная микротрубочка, липидный микроцилиндр, липидная наночастица (LNP) или наноразмерная платформа.
45. Применение антитела по любому из пп. 1-31 или антигенсвязывающего фрагмента по любому из пп. 1-28 в получении лекарственного средства для лечения инфекции SARS-CoV-2 у субъекта.
46. Применение по п. 45, где антитело или антигенсвязывающий фрагмент составлены для введения субъекту внутривенно.
47. Применение по п. 45, где антитело или антигенсвязывающий фрагмент составлены для введения субъекту внутримышечно.
48. Применение по любому из пп. 45-47, где антитело или антигенсвязывающий фрагмент применяют в комбинации с одним или более другими терапевтическими средствами.
49. Применение композиции по пп. 40, 41 или 44 в получении лекарственного средства для лечения инфекции SARS-CoV-2 у субъекта.
50. Применение по п. 49, где композиция составлена для введения субъекту внутривенно.
51. Применение по п. 49, где композиция составлена для введения субъекту внутримышечно.
52. Применение по любому из пп. 49-51, где композицию применяют в комбинации с одним или более другими терапевтическими средствами.
53. Применение по любому из пп. 45-52, где SARS-CoV-2 включает SARS-CoV-2 Wuhan-Hu-1.
54. Способ in vitro получения антитела по любому из пп. 1-31 или антигенсвязывающего фрагмента по любому из пп. 1-28, где способ включает культивирование клетки-хозяина, экспрессирующей антитело или антигенсвязывающий фрагмент, в условиях и в течение времени, достаточных для получения антитела или антигенсвязывающего фрагмента.
55. Способ in vitro по п. 54, где клетка-хозяин содержит рекомбинантный вектор, содержащий выделенный полинуклеотид по любому из пп. 32-36.
56. Способ in vitro по п. 54 или 55, где клетка-хозяин представляет собой клетку млекопитающего.
57. Способ in vitro по любому из пп. 54-56, где способ включает культивирование клетки-хозяина в периодической культуре клеток.
58. Способ in vitro по любому из пп. 54-57, где способ дополнительно включает очистку антитела или антигенсвязывающего фрагмента.
59. Способ лечения инфекции SARS-CoV-2 у субъекта, включающий введение субъекту терапевтически эффективного количества антитела по любому из пп. 1-31 или антигенсвязывающего фрагмента по любому из пп. 1-28 или композиции по любому из пп. 40, 41 и 44.
60. Способ по п. 59, где антитело или антигенсвязывающий фрагмент или композицию вводят субъекту внутривенно.
61. Способ по п. 59, где антитело или антигенсвязывающий фрагмент или композицию вводят субъекту внутримышечно.
62. Способ по любому из пп. 59-61, где SARS-CoV-2 включает SARS-CoV-2 Wuhan-Hu-1.
| XIAOLONG TIAN et al | |||
| Станок для придания концам круглых радиаторных трубок шестигранного сечения | 1924 |
|
SU2019A1 |
| Разборный с внутренней печью кипятильник | 1922 |
|
SU9A1 |
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
| WO 2005012360 A2, 10.02.2005 | |||
| WO 2005060520 A2, 07.07.2005 | |||
| КОМПОЗИЦИИ И СПОСОБЫ ДЛЯ ЛЕЧЕНИЯ ОСТРОГО РЕСПИРАТОРНОГО СИНДРОМА (SARS) | 2004 |
|
RU2411042C2 |
Авторы
Даты
2024-12-18—Публикация
2021-02-25—Подача