Изобретение относится к области генетики, молекулярной биологии, судебной медицины и криминалистики и касается инструмента для установления половой принадлежности, оценки степени деградации ядерной и митохондриальной ДНК и наличия ингибиторов полимеразной цепной реакции (ПЦР) с помощью фрагментного анализа.
Цель предлагаемого способа в том, чтобы проводить мультиплексную ПЦР с последующим фрагментным анализом и «в одной пробирке» оценивать половую принадлежность и степень деградации ДНК сложных биологических образцов, а также определять наличие ингибиторов ПЦР в биоматериале, тем самым устанавливая половую принадлежность, уровень сохранности и загрязненности ингибиторами генетического материала и перспективы его использования для дальнейших более трудоемких методов исследования.
Решение вышеуказанных задач позволяет оптимизировать процесс работы со сложными биологическими объектами, а именно с использованием способа: первично установить половую принадлежность ДНК-образца, определить рациональность использования образца для дальнейших исследований исходя из степени его деградации, а также выбрать тактику ведения исследования, исходя из наличия ингибиторов ПЦР, присутствующих в растворе исследуемой ДНК.
Техническим результатом заявляемого способа является установление половой принадлежности биологического образца, степени деградации ДНК, наличия в нем ингибиторов ПЦР при проведении мультиплексной ПЦР «в одной пробирке» и фрагментного анализа, что позволяет проводить оценку ДНК данного биологического материала для последующего исследования. По результатам фрагментного анализа, исходя из известных ожидаемых длин, специфичных для 4 в случае женской ДНК или 7 в случае мужской ДНК фрагментов, исследуемой ДНК человека и 1 внутреннего положительного контроля (ВПК), по наличию/отсутствию пиков alleledrop-out, их высоте и площади можно первично оценить половую принадлежность ДНК исследуемых образцов по фрагментам генов AmelX/AmelY и SRY, содержащих различной степени деградированный генетический материал, а именно ядерная ДНК – по двум фрагментам гена AmelX/AmelY и митохондриальная ДНК – по фрагменту некодирующего региона HV1 и гена ND1 и ингибиторы ПЦР - по фрагменту гена CSN3B. taurus, представляющему собой ВПК.
В результате за одну реакцию известно три параметра для конкретного препарата ДНК. Учитывая результаты, полученные с использованием описываемого способа, на последующих этапах работы исследователь получает возможность избежать неоправданной траты реактивов и времени.
Генотипирование образцов с различной степенью сохранности генетического материала, включая сложные биологические объекты, содержащие деградированную ДНК: древний костный материал, в том числе обнаруженный в толще почвы; потожировые и иные контактные следы человека; образцы ДНК/крови/тканей, хранящиеся в неблагоприятных условиях, например, на бумажных носителях - ДНК-картах в условиях комнатной температуры; образцы тканей и органов, хранящиеся в водном растворе формальдегида и т. д., представляет собой дорогостоящую и трудозатратную процедуру [1]. Поэтому важным этапом является первичная оценка ДНК исследуемых образцов, содержащих ингибиторы ПЦР, а также различной степени деградированный и генетический материал, позволяющая на следующем этапе исследования из спектра методов подобрать наиболее подходящий, избежав неоправданной траты реактивов и времени.
Новизна и преимущество способа по сравнению с существующими аналогами заключается в возможности более детальной оценки половой принадлежности и степени деградации ДНК по нескольким фрагментам ядерной ДНК, а именно по фрагментам гена амелогенинаХ- и Y-хромосомы и пол-определяющего региона SRY Y-хромосомы, и митохондриальной ДНК- по фрагментам некодирующего региона HV1 и гена ND1, при постановке одной мультиплексной ПЦР-реакции по результатам электрофореза с использованием генетических анализаторов, таких как ABI PRISM3130, ABI PRISM3500, Нанофор-5. Выбраны наиболее распространенные приборы для фрагментного анализа.
Среди методов оценки степени деградации ДНК, половой принадлежности и наличия ингибиторов ПЦР наиболее распространенным являются методы, основанные на применении ПЦР «в реальном времени». Гель-электрофорез выбран в качестве основного инструмента для реализации данного способа в связи с тем, что анализ с его помощью основан на сравнении длины и площади размерных пиков, это позволит более точно в сравнении с методом ПЦР «в реальном времени» оценивать степень деградации, а также устанавливать эффекты выпадения аллелей - allele drop-out.
Важно отметить, что возможность оценки степени деградации митохондриальной ДНК не реализована ни в одном из известных аналогов.
Изначально за аналог принимали набор реактивов «XY-Детект» (Синтол, Россия), снятый в настоящее время с производства. Функционал данного набора реализуется посредством ПЦР «в реальном времени». Предлагается оценка деградации ДНК по 1 короткому фрагменту гена β-актина и более длинному фрагментуX-хромосомы, что не позволяет оценить степень ее деградации детально, это, в свою очередь, накладывает ограничения на точность метода. Также набор не предусматривает оценки степени деградации митохондриальной ДНК; половая принадлежность, аналогично предлагаемому изобретению, устанавливается посредством детекции продуктов амплификации фрагментов амелогенина на Х и Y-хромосомах; наличие ингибиторов ПЦР в растворе ДНК оценивается с помощью ВПК.
В числе российских аналогов со сходным функционалом можно отметить также набор для ПЦР в реальном времени «RealQuantH3» (Синтол, Россия)[2], который предназначен для обнаружения и определения количества ДНК человека, степени ее деградации и половой принадлежности. Помимо перечисленных для предыдущего аналога недостатков, относительно предлагаемого способа, данный продукт не предполагает оценки ингибирования ПЦР.
Для оценки ингибирования ПЦР на российском рынке в настоящее время представлены продукты «А-Детект» (Синтол, Россия; [3]и «Quantum DNA-Set (QS004)» (Евроген, Россия; [4]; позволяет также оценить деградацию ДНК). Оба продукта реализуются посредством ПЦР, что накладывает ограничения на точность процедуры оценки степени ингибирования ПЦР.
Зарубежным аналогом, наиболее приближенным по функционалу к заявляемому способу, является набор реагентов «InvestigatorQuantiplexPro RGQ Kit» (QIAGEN, Germany; [5]. Продукт позволяет оценить половую принадлежность, деградацию ДНК и ингибирование ПЦР, однако не предусматривает оценку деградации митохондриальной ДНК. Существенным отличием также является то, что функционал набора реализуется посредством ПЦР «в реальном времени».
Известен способ по патенту RU 2800083, основанный на использовании системы синтетических олигонуклеотидов для проведения мультиплексной ПЦР с применением для анализа высокопроизводительных технологий, а именно NGS-секвенирования. Недостатками данного способа является невозможность определения половой принадлежности ДНК, а также наличия ингибиторов ПЦР. Несмотря на то, что набор ориентирован на анализ деградированной ДНК - с этой целью праймеры в наборе подобраны таким образцом, чтобы ПЦР-продукт имел минимальную длину меньше 120 п. н., он все же не позволяет определить степень деградации ДНК и, как следствие рациональность ее использования в ходе дальнейших исследований. Набор ориентирован на исследование ядерной ДНК и неприменим к анализу митохондриальной ДНК.
Известен способ по патенту RU 2800084, позволяющий одновременно анализировать множество образцов методом «массового параллельного секвенирования»; данная система олигонуклеотидов адаптирована для использования малых концентраций биологического материала (ДНК) и позволяет определить половую принадлежность ДНК-образца. К недостаткам данного метода относятся невозможность определения половой принадлежности ДНК, наличия ингибиторов ПЦР, а также ориентированность на работу лишь с ядерной ДНК.
Известен способ по патенту CN110055337A, позволяющий определить половую принадлежность древних человеческих останков с очень низким содержанием ДНК. Способ, аналогично предлагаемому в данном изобретении, предполагает проведение ПЦР с использованием флуоресцентно меченных олигонуклеотидов с последующим гель-электрофорезом. Однако способ не предполагает установление степени деградации ДНК и наличия ингибиторов ПЦР.
Таким образом, готового продукта или способа, позволяющего комплексно на количественном уровне оценить пригодность ДНК-образца, в том числе древнего ДНК-образца, с учетом половой принадлежности, степени его деградации и наличия ингибиторов ПЦР в препарате ДНК, к последующему анализу с использованием дорогих и трудозатратных методик до настоящего времени предложено не было.
Описание
Технической задачей предполагаемого изобретения является разработка комплексного способа установления половой принадлежности, оценки степени деградации ядерной и митохондриальной ДНК и наличия ингибиторов ПЦР посредством фрагментного анализа.
Поставленная задача достигается тем, что способ установления половой принадлежности, оценки степени деградации ядерной и митохондриальной ДНК и наличия ингибиторов ПЦР включает выделение ДНК из исследуемого материала, постановку ПЦР со специфичными праймерами, несущими флуоресцентную метку, и учет полученных данных по результатам фрагментного анализа полученных в ходе ПЦР ампликонов.
Основными критериями подбора пар праймеров для реализации заявленного способа служат:
1. Для оценки половой принадлежности ДНК человека используются праймеры, места отжига которых фланкируют делеции гена AmelX. За счет этого продукты амплификации данных фрагментов гена амелогенина будут отличаться для Х- и Y-хромосомы, что позволит их безошибочно идентифицировать и, тем самым, установить половую принадлежность индивидуума.
2. Для оценки степени деградации ядерной ДНК человека используются две пары праймеров на два фрагмента гена амелогенина разной длины; для оценки степени деградации митохондриальной ДНК человека используются праймеры на два фрагмента митохондриона человека разной длины.
3. Для оценки наличия ингибиторов ПЦР используется пара праймеров на фрагмент гена CSN3 B. taurus, служащего ВПК протекания реакции.
4. Все используемые пары праймеров должны иметь высокую степень специфичности к заявляемым участкам генома.
Таким образом, амплификацию исследуемой ДНК проводят с помощью предлагаемых пар праймеров (последовательности праймеров представлены в Таб. 1), имеющих специфичную длину для каждого исследуемого фрагмента ДНК и несущих флуоресцентную метку FAM на 5’-конце прямого праймера, что позволяет реализовать заявленные для способа функции посредством фрагментного анализа. Часть праймеров сгенерирована самостоятельно с помощью ресурса NCBIBlast, а часть – взята из литературных источников.
Таблица 1. Характеристики праймеров, используемых для реализации способа
*Длина продукта может отличаться от теоретической вследствие особенностей биоинформационной обработки результатов фрагментного анализа
**Праймеры специфичны гену CSN3 B. taurus
***Праймеры используются впервые, были сгенерированы авторами способа с помощью ресурса NCBI Blast
Несмотря на то, что в заявляемом изобретении в качестве меток специфичных олигонуклеотидов используется метка FAM, вместо нее может использоваться любая другая флуоресцирующая метка, что никак не изменит заявленного функционала изобретения, как следствие, модификации такого рода также будут входить в объем притязаний настоящего изобретения.
ПЦР смесь, в зависимости от необходимого функционала способа, должна включать компоненты, указанные в Таблице 2.
Таблица 2. Состав ПЦР-смеси в зависимости от необходимого функционала способа
(несут флуоресцентную метку FAM на 5’-конце)
1. Установление половой принадлежности
Установление половой принадлежности ДНК-образца является рутинной процедурой в области судебной медицины и криминалистики. Процесс определения половой принадлежности на основе делетированных областей гена амелогенина уже известен [6]. Оптимизация процесса состоит в использовании в реакции одновременно двух пар праймеров на короткий и длинный фрагменты гена амелогенина и пары праймеров на короткий фрагмент гена SRY. Использование двух пар праймеров позволит с большей вероятностью, чем при использовании одной пары праймеров, безошибочно определить половую принадлежность ДНК, учитывая случаи сильной деградации ДНК, при которой возможно выпадение фрагмента на Y-хромосоме, приводящее к ложному определению пола. Пара праймеров на короткий фрагмент гена SRY служит в качестве дополнительного контроля половой принадлежности в случае «выпадения» фрагментов AmelY, специфичных для мужского пола.
Половую принадлежность предполагается устанавливать посредством учета разницы длин продуктов амплификации гена амелогенина для X и Y хромосом. Разница длин фрагментов для данного гена обусловлена наличием делетированных областей на Х-хромосоме. Так, в случае Х-хромосомы детектируются продукты длиной 80 п.н.- в случае короткого и 212 п.н. – в случае длинного фрагмента гена AmelX. Для Y-хромосомы детектируются пики длиной 83 п.н. - в случае короткого и 218 п.н. – в случае длинного фрагмента гена AmelY.
Фрагмент гена SRY длиной 93 п.н. является маркером наличия в генотипе человека Y-хромосомы и служит в качестве дополнительного контроля половой принадлежности в случае «выпадения» фрагментов AmelY, специфичных для мужского пола.
2. Оценка степени деградации ядерной ДНК
Геном человека включает в себя ядерную и митохондриальную ДНК. В ходе криминалистических исследований ядерная ДНК в основном используется для STR-анализа, а митохондриальная ДНК, наследуясь исключительно по материнской линии, является инструментом для установления родства и, учитывая вариабельность участков D-петли, популяционных характеристик.
Степень деградации ядерной и митохондриальной ДНК является важным показателем, позволяющим оценить пригодность использования конкретного ДНК-препарата в исследованиях с применением вышеуказанных молекулярно-генетических инструментов. Для оценки степени деградации ДНК в настоящее время применяется метод с использованием ПЦР «в реальном времени», основанный на определении степени фрагментации ДНК по соотношению эффективных концентраций короткого и длинного фрагментов. Однако такой метод не позволяет на необходимом количественном уровне оценить степень деградации ДНК. В связи с чем процесс оценки в случае описываемого метода предлагается осуществлять посредством фрагментного анализа.
Так, исходя из цели конкретного исследования может возникать необходимость использования тест-систем и наборов реактивов, реализуемых через применение праймеров на фрагменты генов разной длины. Понимание степени деградации конкретного препарата ДНК позволит спрогнозировать вероятность отжига праймеров определенной длины и, как следствие рациональность его использования для исследований.
Например, проведение молекулярно-генетического STR-анализа с использованием наборов реактивов «COrDIS» (Гордиз, Россия) предполагает использование наборов праймеров на фрагменты разной длины: от 81 до 450 п.н. Предварительное применение разработанного нами метода позволит спрогнозировать выпадение фрагментов и определить рациональность траты реактивов для исследования конкретного ДНК-образца.
Оценку степень деградации ДНК предполагается проводить посредством использования двух пар праймеров на два фрагмента ядерной ДНК (ген AmelX/AmelY) разной длины – короткий 80-83 п.н. и длинный 212-218 п.н. Определение степени деградации митохондриальной ДНК не реализовано ни в одном из известных патентов или коммерческих предложений. С этой целью предлагается использование пары праймеров на короткий фрагмент участка HV1 длиной 137 п.н. и пары праймеров на длинный фрагмент гена ND1 длиной 235 п.н.
В случае, когда при использовании способа на электрофореграмме кроме пика, соответствующего ВПК, другие пики не детектируются, можно сделать вывод о нерациональности проведения анализа для конкретного ДНК-образца в связи с полной деградацией генетического материала человека.
3. Наличие ингибиторов ПЦР
Ингибирование ПЦР является серьезной проблемой в молекулярной биологии препятствующей протеканию реакции ПЦР, без которой, по сути, невозможна реализация методов секвенирования и фрагментного анализа.
Оценка наличия ингибиторов ПЦР в ДНК-образце будет производиться за счет наличия в ПЦР-смеси ВПК – нечеловеческой ДНК (Bostaurus) и пары специфичных праймеров на фрагмент гена CSN3 длиной 351 п.н. Показатель степени ингибирования ПЦР рассчитывается для опытной пробы относительно контрольного образца по значениям RFU для пика, соответствующим ожидаемой длине 351 п.н. в процентном выражении и служит показателем, на основании которого исследователь имеет возможность при необходимости самостоятельно реализовать поиск путей преодоления ингибирования ПЦР.
4. Анализ результатов фрагментного анализа
Анализ результатов фрагментного анализа будет производиться автоматически посредством соответствующего программного обеспечения, например GeneMapper, GeneMapperID (IDx), GeneMarker. На основании полученных пиков для фрагментов различной длины исследователем делается вывод о половой принадлежности, степени деградации ДНК-образца и наличии ингибиторов ПЦР.
Краткое описание иллюстраций, демонстрирующих сущность заявляемого способа:
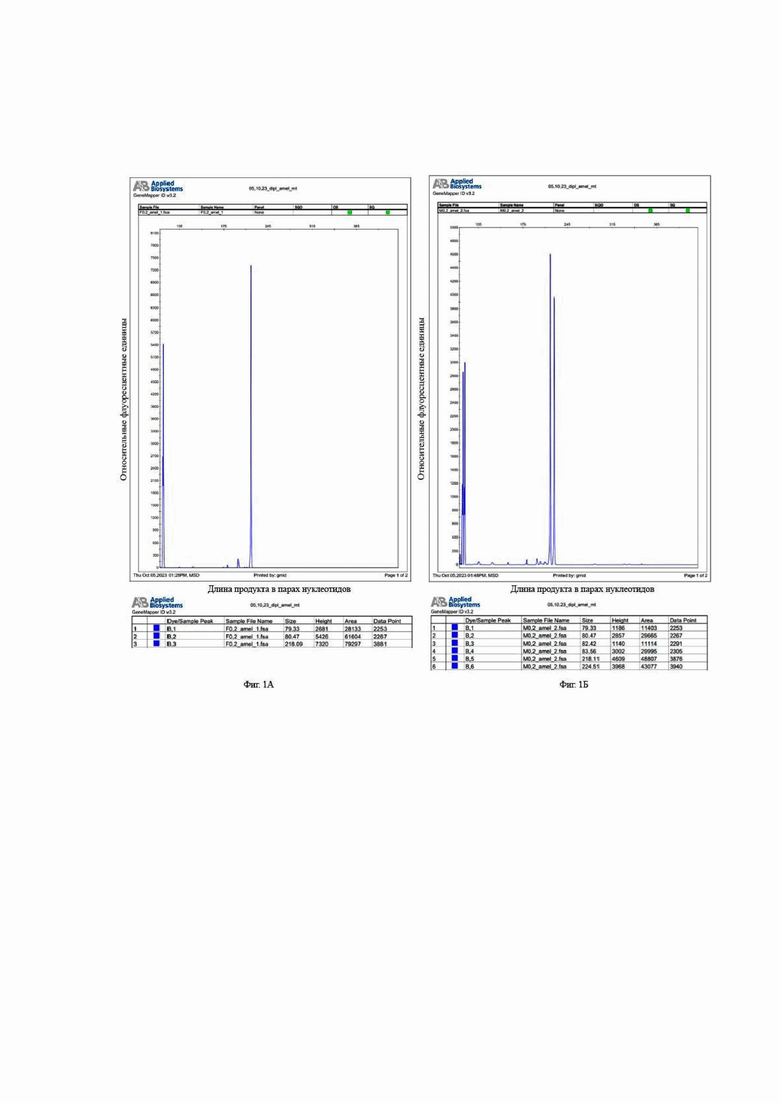
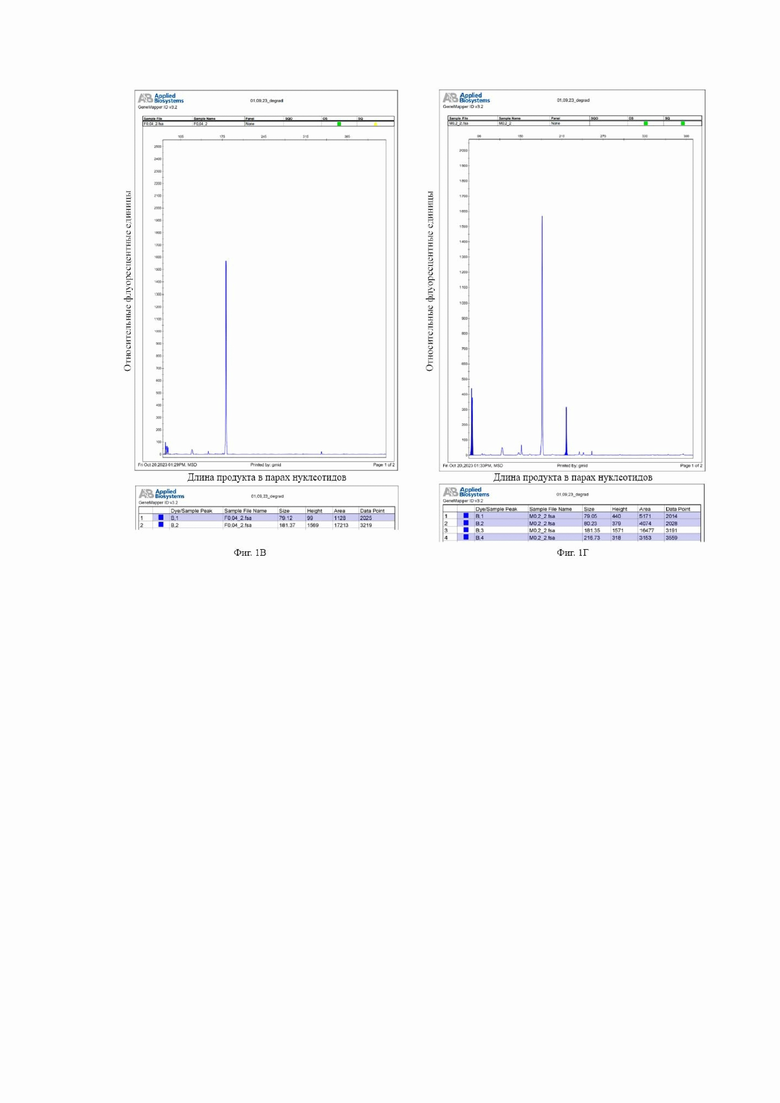
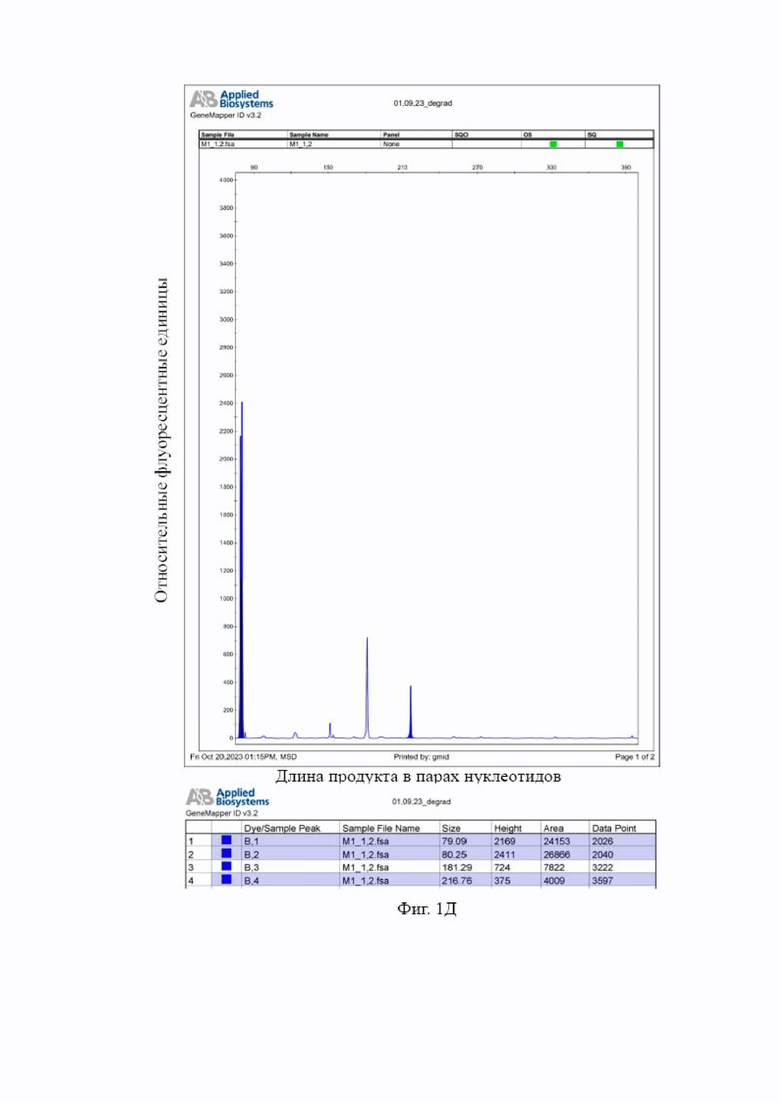
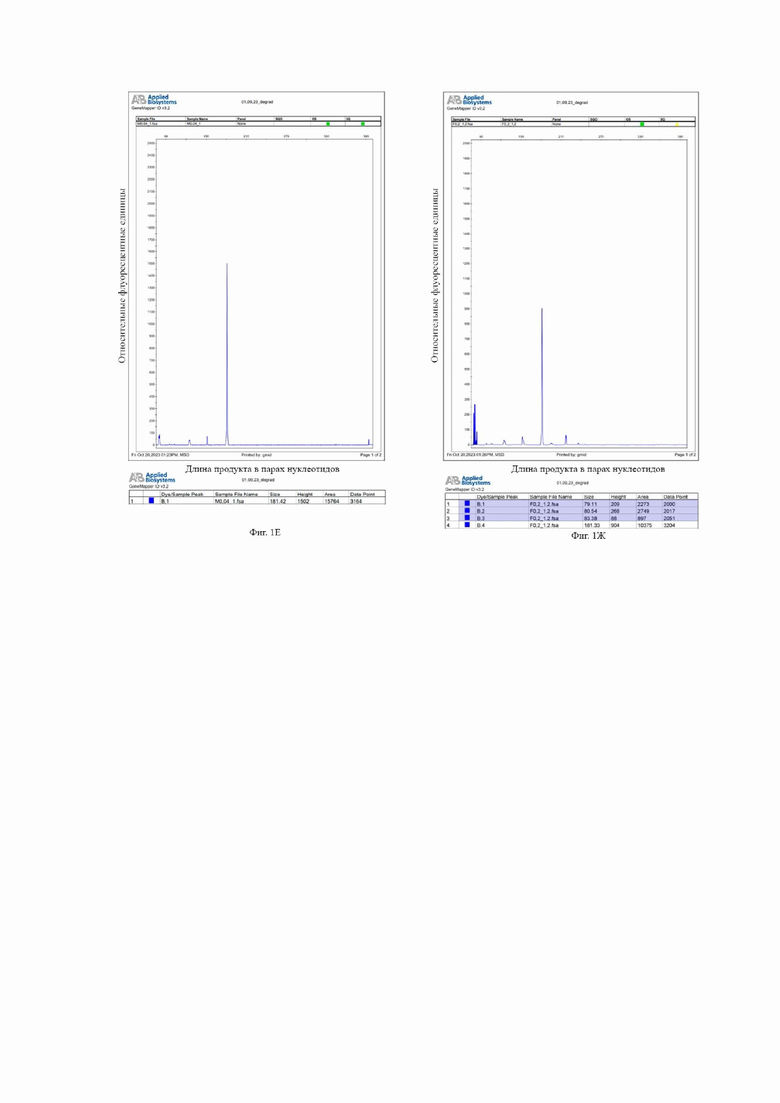
На Фиг. 1 представлены результаты фрагментного анализа ампликонов, полученных в ходе дуплексной ПЦР-реакции с использованием двух пар праймеров Amel_212-218_F/Amel_212-218_R и Amel_80-83_F/Amel_80-83_R, с целью определения половой принадлежности и степени деградации ядерной ДНК: на Фиг 1А – для недеградированной женской ДНК в начальной концентрации 0,2 нг/мкл; на Фиг 1Б – для недеградированной мужской ДНК в начальной концентрации 0,2 нг/мкл; на Фиг 1В – для деградированной женской ДНК в начальной концентрации 0,04 нг/мкл; на Фиг 1Г – для деградированной женской ДНК в начальной концентрации 0,2 нг/мкл; на Фиг 1Д – для деградированной женской ДНК в начальной концентрации 1 нг/мкл; на Фиг 1Е– для деградированной мужской ДНК в начальной концентрации 0,04 нг/мкл; на Фиг 1Ж – для деградированной мужской ДНК в начальной концентрации 0,2 нг/мкл; на Фиг 1З – для деградированной мужской ДНК в начальной концентрации 1 нг/мкл.
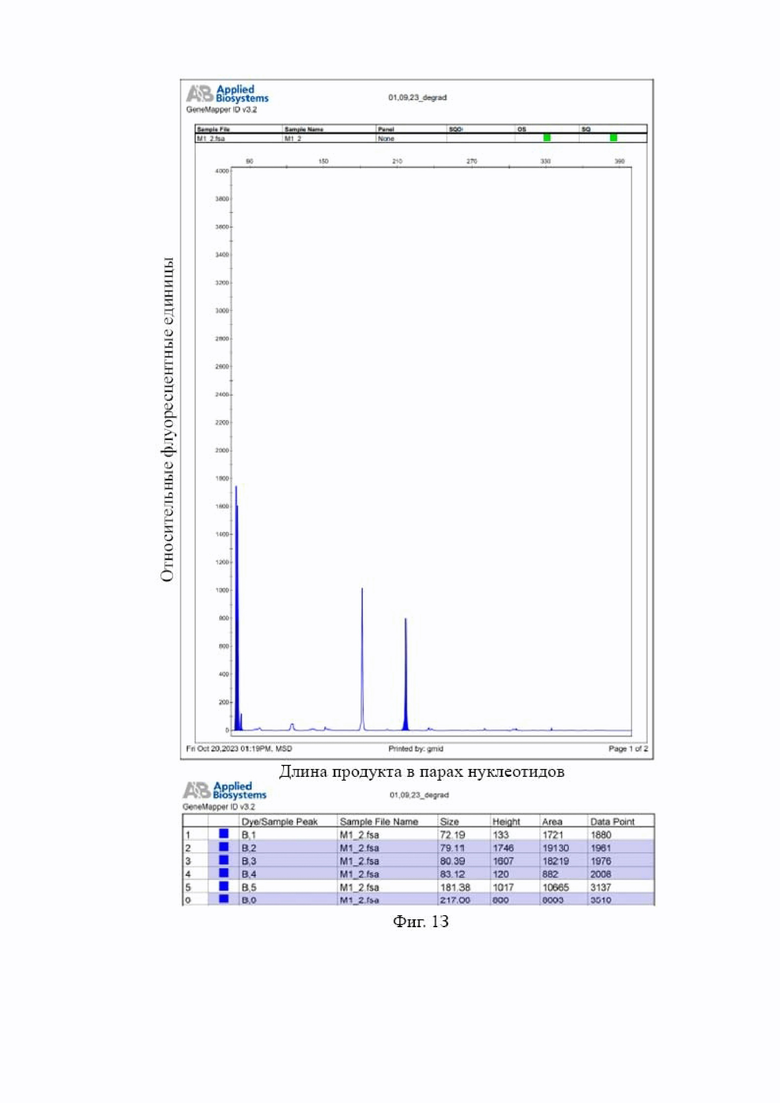
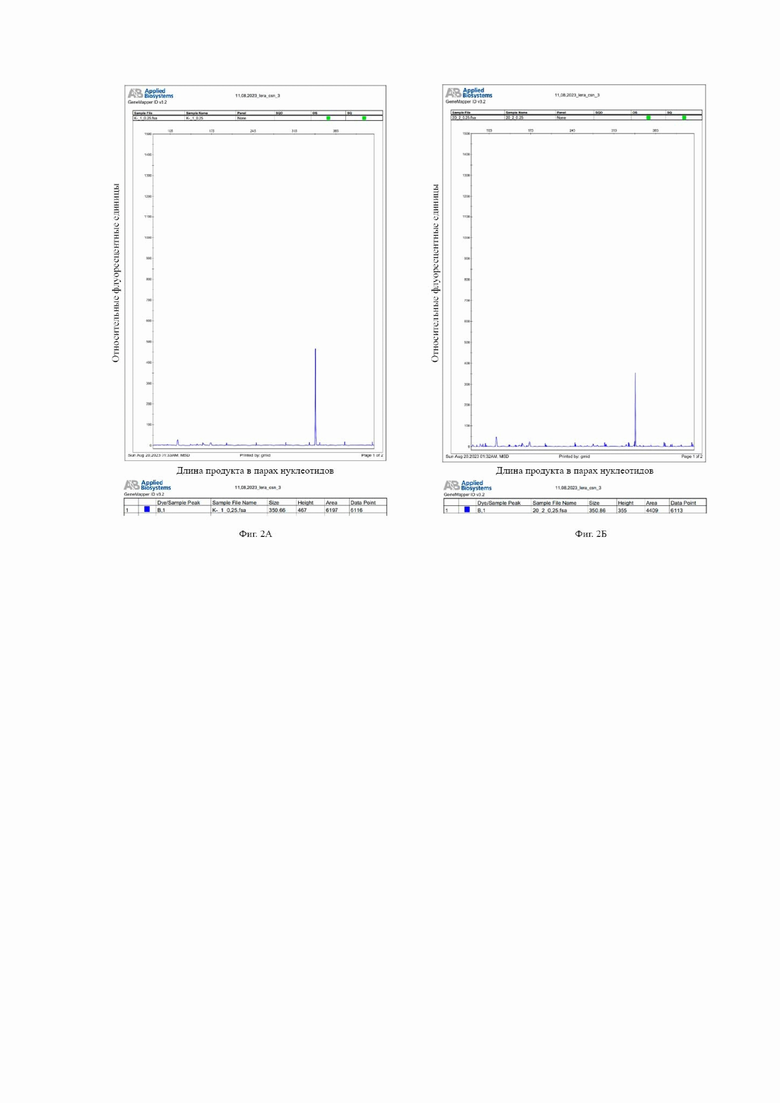
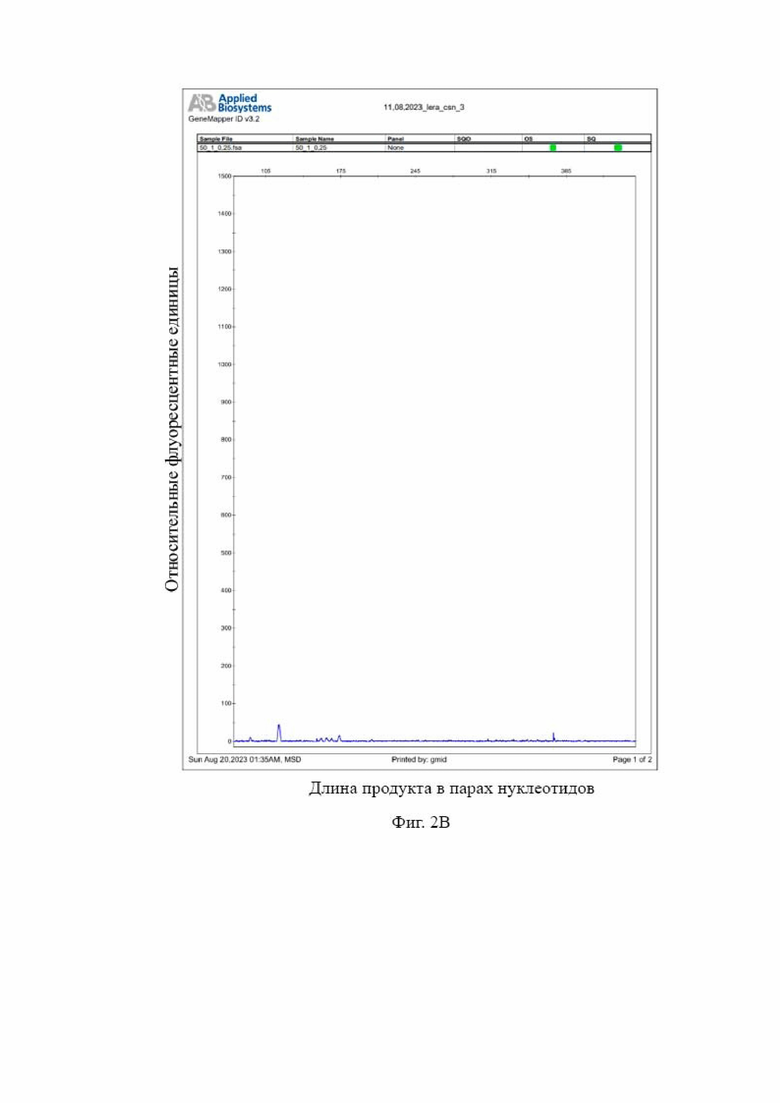
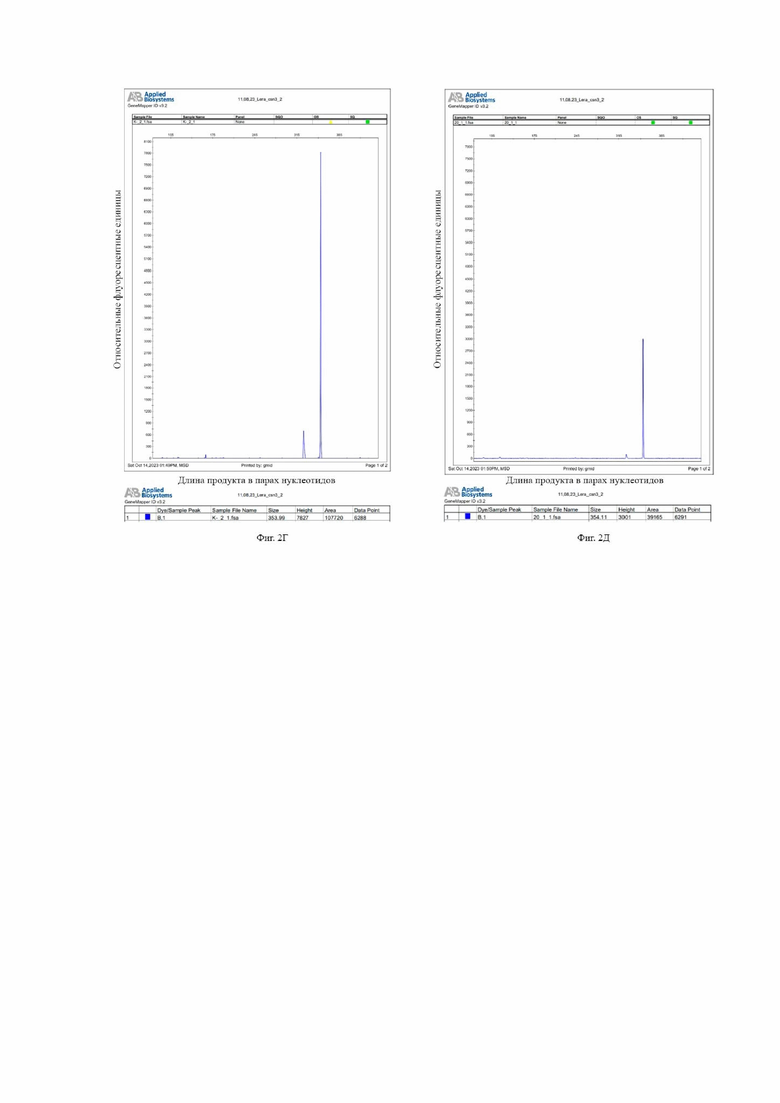
На Фиг. 2 представлены результаты фрагментного анализа ампликонов, полученных в ходе ПЦР-реакции с использованием одной пары праймеров CSN3_351_F/CSN3_351_R на участок гена CSN3 B. taurus (в качестве ВПК реакции) с целью определения наличия ингибиторов ПЦР: на Фиг 2А – для ДНК B. taurus в конечной концентрации0,01 нг/мкл, вместо ФК использовали дистиллированную воду; на Фиг 2Б – для ДНК B. taurus в конечной концентрации 0,01 нг/мкл, ФК в конечной концентрации 20 нг/мкл; на Фиг 2В – для ДНК B. taurus в конечной концентрации 0,01 нг/мкл, ФК в конечной концентрации 50 нг/мкл; на Фиг 2Г – для ДНК B. taurus в конечной концентрации 0,04 нг/мкл, вместо ФК использовали дистиллированную воду; на Фиг 2Д – для ДНК B. taurus в конечной концентрации 0,04 нг/мкл, ФК в конечной концентрации 20 нг/мкл; на Фиг 2Е – для ДНК B. taurus в конечной концентрации 0,04 нг/мкл, ФК в конечной концентрации 50 нг/мкл.
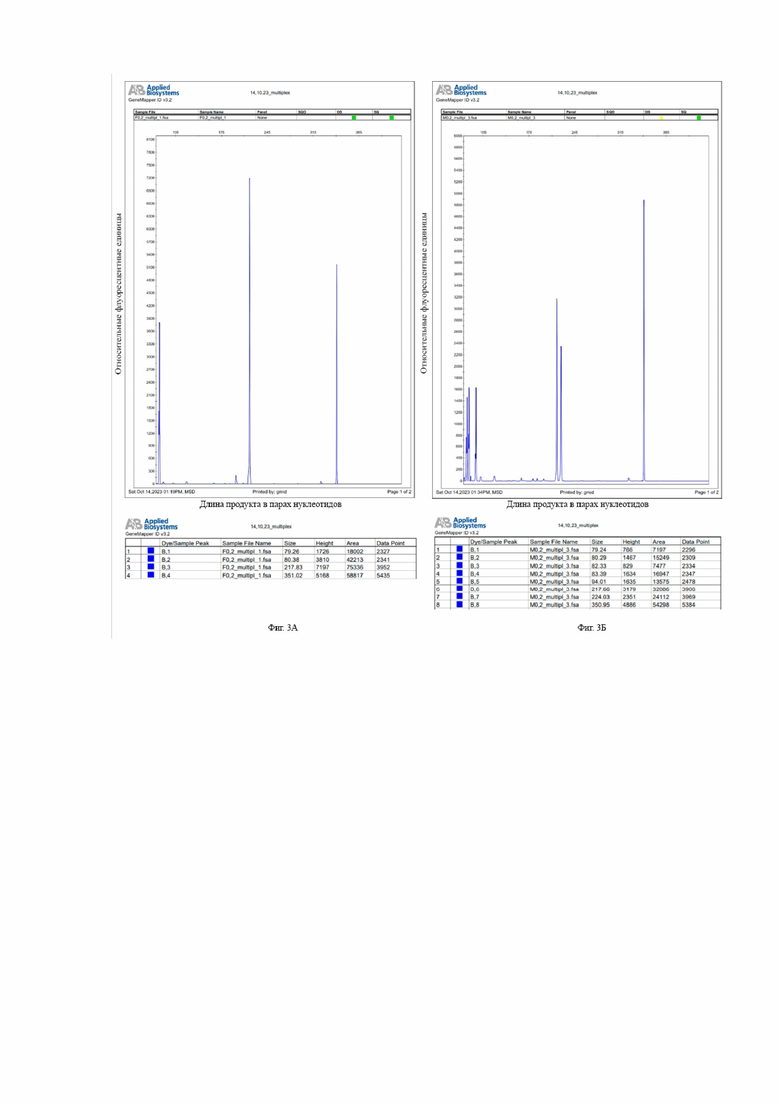
На Фиг. 3 представлены результаты фрагментного анализа ампликонов, полученных в ходе мультиплексной ПЦР-реакции с использованием четырех пар праймеров: Amel_212-218_F/Amel_212-218_R, Amel_80-83_F/Amel_80-83_R,SRY_93_F/SRY_93_R (на фрагменты генов H. sapiens), CSN3_351_F/CSN3_351_R (на фрагмент гена B. taurus), с целью определения половой принадлежности, степени деградации ядерной ДНК и наличия ингибиторов ПЦР: на Фиг 3А - для недеградированной женской ДНК в начальной концентрации 0,2 нг/мкл; на Фиг. 3Б – для недеградированной мужской ДНК в начальной концентрации 0,2 нг/мкл; на Фиг. 3В – для деградированной женской ДНК в начальной концентрации 0,2 нг/мкл; на Фиг 3Г – для деградированной мужской ДНК в начальной концентрации 0,2 нг/мкл.
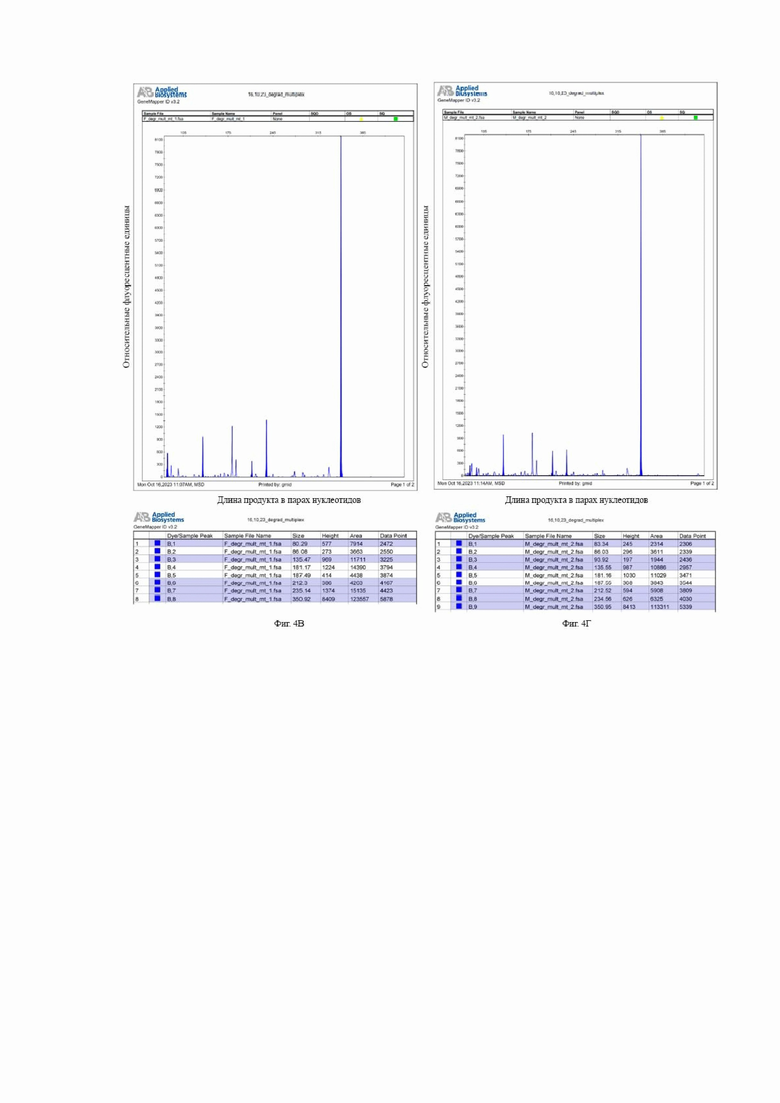
На Фиг. 4 представлены результаты фрагментного анализа ампликонов, полученных в ходе мультиплексной ПЦР-реакции с использованием шести пар праймеров: Amel_212-218_F/Amel_212-218_R, Amel_80-83_F/Amel_80-83_R,SRY_93_F/SRY_93_R,ND1_235_F/ND1_235_R;HV1_137_F/HV1_137_R (на фрагменты генов H. sapiens), CSN3_351_F/CSN3_351_R (на фрагмент гена B. taurus), с целью определения половой принадлежности, степени деградации ядерной и митохондриальной ДНК и наличия ингибиторов ПЦР: на Фиг 4А - для недеградированной женской ДНК в начальной концентрации 0,2 нг/мкл; на Фиг. 4Б – для недеградированной мужской ДНК в начальной концентрации 0,2 нг/мкл; на Фиг. 4В – для деградированной женской ДНК в начальной концентрации 0,2 нг/мкл; на Фиг 4Г – для деградированной мужской ДНК в начальной концентрации 0,2 нг/мкл.
Осуществление способа
Формула 1
Способ установления половой принадлежности, оценки степени деградации ядерной и митохондриальной ДНК и наличия ингибиторов ПЦР с помощью фрагментного анализа, который включает на первом этапе проведение дуплексной полимеразной цепной реакции с использованием ПЦР-смеси, включающей:
10-кратный ПЦР буфер – 2,5 мкл:
2,5 мМ смесь dNTP – 2,5 мкл;
25 мМ MgCl2 – 1,5-2,5 мкл;
ДНК-Taq-полимеразу 5 Ед/мкл – 0,3-0,7 мкл;
пару праймеров Amel_212-218_F/Amel_212-218_R – по 0,1-1 о.е./мкл каждого праймера;
пару праймеров Amel_80-83_F/Amel_80-83_R– по 0,5-2 о.е./мкл каждого праймера;
исследуемый образец ДНК – 2,5 мкл в начальной концентрации 0,1-1 нг/мкл;
дистиллированную воду – до конечного объема реакционной смеси 25 мкл;
на втором этапе осуществляют анализ полученных ампликонов на ДНК-анализаторе методом капиллярного гель-электрофореза – фрагментный анализ.
Формула 2
Способ по п.1 для установления половой принадлежности и оценки степени деградации ядерной ДНК человека, где в качестве дополнительного контроля пола индивида используется также праймеров на фрагмент гена SRY, т.о. ПЦР-смесь содержит:
10-кратный ПЦР буфер – 2,5 мкл:
2,5 мМ смесь dNTP – 2,5 мкл;
25 мМ MgCl2 – 1,5-2,5 мкл;
ДНК-Taq-полимеразу 5 Ед/мкл – 0,3-0,7 мкл;
пару праймеров Amel_212-218_F/Amel_212-218_R – по 0,1-1 о.е./мкл каждого праймера;
пару праймеров Amel_80-83_F/Amel_80-83_R – по 0,5-2 о.е./мкл каждого праймера;
пару праймеров SRY_93_F/SRY_93_R – по 0,1-1,5 о.е/мкл каждого праймера;
исследуемый образец ДНК – 2,5 мкл в начальной концентрации 0,1-1 нг/мкл;
дистиллированную воду – до конечного объема реакционной смеси 25 мкл;
на втором этапе осуществляют анализ полученных ампликонов на ДНК-анализаторе методом капиллярного гель-электрофореза – фрагментный анализ.
Формула 3
Способ для определения наличия ингибиторов ПЦР, который включает на первом этапе проведение полимеразной цепной реакции с использованием ПЦР-смеси, содержащей ДНК данного организма в начальной концентрации 0,25-1 нг/мкл, включающей одну специфичную пару праймеров на фрагмент гена CSN3 домашнего крупного рогатого скота Bostaurus:
10-кратный ПЦР буфер – 2,5 мкл:
2,5 мМ смесь dNTP – 2,5 мкл;
25 мМ MgCl2 – 1,5-2,5 мкл;
ДНК-Taq-полимеразу 5 Ед/мкл – 0,3-0,7 мкл;
пару праймеров CSN3_351_F/CSN3_351_R – по 0,5-2 о.е./мкл каждого праймера;
ДНК быка – 1 мкл в начальной концентрации 0,25-1 нг/мкл;
дистиллированную воду – до конечного объема реакционной смеси 25 мкл;
на втором этапе осуществляют анализ полученных ампликонов на ДНК-анализаторе методом капиллярного гель-электрофореза – фрагментный анализ.
Формула 4
Способ по пп. 1-3 для установления половой принадлежности, оценки степени деградации ядерной ДНК человека и наличия ингибиторов ПЦР, т.о. состав ПЦР-смеси для проведения триплексной реакции включает:
10-кратный ПЦР буфер – 2,5 мкл:
2,5 мМ смесь dNTP – 2,5 мкл;
25 мМ MgCl2 – 1,5-2,5 мкл;
ДНК-Taq-полимеразу 5 Ед/мкл – 0,3-0,7 мкл;
пару праймеров Amel_212-218_F/Amel_212-218_R – по 0,1-1 о.е./мкл каждого праймера;
пару праймеров Amel_80-83_F/Amel_80-83_R – по 0,5-2 о.е./мкл каждого праймера;
пару праймеров CSN3_351_F/CSN3_351_R – по 0,5-2 о.е./мкл каждого праймера;
ДНК быка – 1 мкл в начальной концентрации 0,25-1 нг/мкл;
исследуемый образец ДНК – 2,5 мкл в начальной концентрации 0,1-1 нг/мкл;
дистиллированную воду – до конечного объема реакционной смеси 25 мкл;
на втором этапе осуществляют анализ полученных ампликонов на ДНК-анализаторе методом капиллярного гель-электрофореза – фрагментный анализ.
Формула 5
Способ по п. 4 для установления половой принадлежности, где в качестве дополнительного контроля пола индивида используется также пара праймеров на фрагмент гена SRY, оценки степени деградации ядерной ДНК человека и наличия ингибиторов ПЦР, т.о. состав ПЦР-смеси для проведения мультиплексной реакции включает:
10-кратный ПЦР буфер – 2,5 мкл:
2,5 мМ смесь dNTP – 2,5 мкл;
25 мМ MgCl2 – 1,5-2,5 мкл;
ДНК-Taq-полимеразу 5 Ед/мкл – 0,3-0,7 мкл;
пару праймеров Amel_212-218_F/Amel_212-218_R – по 0,1-1 о.е./мкл каждого праймера;
пару праймеров Amel_80-83_F/Amel_80-83_R – по 0,5-2 о.е./мкл каждого праймера;
пару праймеров SRY_93_F/SRY_93_R – по 0,1-1,5 о.е/мкл каждого праймера;
пару праймеров CSN3_351_F/CSN3_351_R – по 0,5-2 о.е./мкл каждого праймера;
ДНК быка – 1 мкл в начальной концентрации 0,25-1 нг/мкл;
исследуемый образец ДНК – 2,5 мкл в начальной концентрации 0,1-1 нг/мкл;
дистиллированную воду – до конечного объема реакционной смеси 25 мкл;
на втором этапе осуществляют анализ полученных ампликонов на ДНК-анализаторе методом капиллярного гель-электрофореза – фрагментный анализ.
Формула 6
Способ для оценки степени деградации митохондриальной ДНК человека, который включает на первом этапе проведение дуплексной полимеразной цепной реакции с использованием ПЦР-смеси, включающей:
10-кратный ПЦР буфер – 2,5 мкл:
2,5 мМ смесь dNTP – 2,5 мкл;
25 мМ MgCl2 – 1,5-2,5 мкл;
ДНК-Taq-полимеразу 5 Ед/мкл – 0,3-0,7 мкл;
пару праймеров ND1_235_F/ND1_235_R – по 0,05-1 о.е./мкл каждого праймера;
пару праймеров HV1_137_F/HV1_137_R – по 0,1-1 о.е./мкл каждого праймера;
исследуемый образец ДНК – 2,5 мкл в начальной концентрации 0,1-1 нг/мкл;
дистиллированную воду – до конечного объема реакционной смеси 25 мкл;
на втором этапе осуществляют анализ полученных ампликонов на ДНК-анализаторе методом капиллярного гель-электрофореза – фрагментный анализ.
Формула 7
Способ по п. 3 и п. 6 для оценки степени деградации митохондриальной ДНК человека и наличия ингибиторов ПЦР, т.о. в триплексную реакцию включены:
10-кратный ПЦР буфер – 2,5 мкл:
2,5 мМ смесь dNTP – 2,5 мкл;
25 мМ MgCl2 – 1,5-2,5 мкл;
ДНК-Taq-полимераза 5 Ед/мкл – 0,3-0,7 мкл;
пара праймеров ND1_235_F/ND1_235_R – по 0,05-1 о.е./мкл каждого праймера;
пара праймеров HV1_137_F/HV1_137_R – по 0,1-1 о.е./мкл каждого праймера;
пара праймеров CSN3_351_F/CSN3_351_R – по 0,5-2 о.е./мкл каждого праймера;
ДНК быка – 1 мкл в начальной концентрации 0,25-1 нг/мкл;
исследуемый образец ДНК – 2,5 мкл в начальной концентрации 0,1-1 нг/мкл;
дистиллированная вода – до конечного объема реакционной смеси 25 мкл;
на втором этапе осуществляют анализ полученных ампликонов на ДНК-анализаторе методом капиллярного гель-электрофореза – фрагментный анализ.
Формула 8
Способ по п. 1 и п. 6 для оценки степени деградации ядерной и митохондриальной ДНК человека, т.о. в мультиплексную реакцию включены:
10-кратный ПЦР буфер – 2,5 мкл:
2,5 мМ смесь dNTP – 2,5 мкл;
25 мМ MgCl2 – 1,5-2,5 мкл;
ДНК-Taq-полимераза 5 Ед/мкл – 0,3-0,7 мкл;
пара праймеров Amel_212-218_F/Amel_212-218_R – по 0,1-1 о.е./мкл каждого праймера;
пара праймеров Amel_80-83_F/Amel_80-83_R – по 0,5-2 о.е./мкл каждого праймера;
пара праймеров ND1_235_F/ND1_235_R – по 0,05-1 о.е./мкл каждого праймера;
пара праймеров HV1_137_F/HV1_137_R – по 0,1-1 о.е./мкл каждого праймера;
исследуемый образец ДНК – 2,5 мкл в начальной концентрации 0,1-1 нг/мкл;
дистиллированная вода – до конечного объема реакционной смеси 25 мкл;
на втором этапе осуществляют анализ полученных ампликонов на ДНК-анализаторе методом капиллярного гель-электрофореза – фрагментный анализ.
Формула 9
Способ по п. 3 и п. 8 для установления половой принадлежности, оценки степени деградации ядерной и митохондриальной ДНК человека и наличия ингибиторов ПЦР, т.о. в мультиплексную реакцию включены:
10-кратный ПЦР буфер – 2,5 мкл:
2,5 мМ смесь dNTP – 2,5 мкл;
25 мМ MgCl2 – 1,5-2,5 мкл;
ДНК-Taq-полимераза 5 Ед/мкл – 0,3-0,7 мкл;
пара праймеров Amel_212-218_F/Amel_212-218_R – по 0,1-1 о.е./мкл каждого праймера;
пара праймеров Amel_80-83_F/Amel_80-83_R – по 0,5-2 о.е./мкл каждого праймера;
пара праймеров ND1_235_F/ND1_235_R – по 0,05-1 о.е./мкл каждого праймера;
пара праймеров HV1_137_F/HV1_137_R – по 0,1-1 о.е./мкл каждого праймера;
пара праймеров CSN3_351_F/CSN3_351_R – по 0,5-2 о.е./мкл каждого праймера;
ДНК быка – 1 мкл в начальной концентрации 0,25-1 нг/мкл;
исследуемый образец ДНК – 2,5 мкл в начальной концентрации 0,1-1 нг/мкл;
дистиллированная вода – до конечного объема реакционной смеси 25 мкл;
на втором этапе осуществляют анализ полученных ампликонов на ДНК-анализаторе методом капиллярного гель-электрофореза – фрагментный анализ.
Формула 10
Способ по п. 2 и п. 6 для установления половой принадлежности, оценки степени деградации ядерной, где в качестве дополнительного контроля пола индивида используется также пара праймеров на фрагмент гена SRY, и митохондриальной ДНК человека, т.о. в мультиплексную реакцию включены:
10-кратный ПЦР буфер – 2,5 мкл:
2,5 мМ смесь dNTP – 2,5 мкл;
25 мМ MgCl2 – 1,5-2,5 мкл;
ДНК-Taq-полимераза 5 Ед/мкл – 0,3-0,7 мкл;
пара праймеров Amel_212-218_F/Amel_212-218_R – по 0,1-1 о.е./мкл каждого праймера;
пара праймеров Amel_80-83_F/Amel_80-83_R – по 0,5-2 о.е./мкл каждого праймера;
пара праймеров SRY_93_F/SRY_93_R – по 0,1-1,5 о.е/мкл каждого праймера;
пара праймеров ND1_235_F/ND1_235_R – по 0,05-1 о.е./мкл каждого праймера;
пара праймеров HV1_137_F/HV1_137_R – по 0,1-1 о.е./мкл каждого праймера;
исследуемый образец ДНК – 2,5 мкл в начальной концентрации 0,1-1 нг/мкл;
дистиллированная вода – до конечного объема реакционной смеси 25 мкл;
на втором этапе осуществляют анализ полученных ампликонов на ДНК-анализаторе методом капиллярного гель-электрофореза – фрагментный анализ.
Формула 11
Способ по п. 3 и п. 10 для установления половой принадлежности, оценки степени деградации ядерной, где в качестве дополнительного контроля пола индивида используется также пара праймеров на фрагмент гена SRY, и митохондриальной ДНК человека и наличия ингибиторов ПЦР, т.о. в мультиплексную реакцию включены:
10-кратный ПЦР буфер – 2,5 мкл:
2,5 мМ смесь dNTP – 2,5 мкл;
25 мМ MgCl2 – 1,5-2,5 мкл;
ДНК-Taq-полимераза 5 Ед/мкл – 0,3-0,7 мкл;
пара праймеров Amel_212-218_F/Amel_212-218_R – по 0,1-1 о.е./мкл каждого праймера;
пара праймеров Amel_80-83_F/Amel_80-83_R – по 0,5-2 о.е./мкл каждого праймера;
пара праймеров SRY_93_F/SRY_93_R – по 0,1-1,5 о.е/мкл каждого праймера;
пара праймеров ND1_235_F/ND1_235_R – по 0,05-1 о.е./мкл каждого праймера;
пара праймеров HV1_137_F/HV1_137_R – по 0,1-1 о.е./мкл каждого праймера;
пара праймеров CSN3_351_F/CSN3_351_R – по 0,5-2 о.е./мкл каждого праймера;
ДНК быка – 1 мкл в начальной концентрации 0,25-1 нг/мкл;
исследуемый образец ДНК – 2,5 мкл в начальной концентрации 0,1-1 нг/мкл;
дистиллированная вода – до конечного объема реакционной смеси 25 мкл;
на втором этапе осуществляют анализ полученных ампликонов на ДНК-анализаторе методом капиллярного гель-электрофореза – фрагментный анализ.
Сущность изобретения поясняется следующими примерами, которые показывают применение способа установления половой принадлежности, оценки степени деградации ядерной и митохондриальной ДНК и наличия ингибиторов ПЦР. Следует понимать, что приводимые примеры служат для демонстрации вариативности применения заявляемого способа.
Пример № 1
С целью иллюстрации способа определения половой принадлежности и степени деградации ядерной ДНК осуществляли выделение ДНК из клеток буккального эпителия, полученного от двух лиц (мужского и женского пола), путемфенол-органической экстракции[1].Для дополнительной очистки и концентрирования препарата ДНК использовали устройство «AmiconUltra-4, ultracel30k» (Merck, Германия).
Концентрацию и чистоту полученных препаратов ДНК оценивали спектрофотометрически, регистрируя ультрафиолетовые спектры с помощью спектрофотометра ND-1000 (NanoDropTechnologies, США).
Препараты ДНК нормировали до конечной концентрации 0,2 нг/мкл с помощью стерильной деионизованной воды. Полученные образцы мужской и женской ДНК считали материалом для иллюстрации работы способа с недеградированной ДНК.
Для постановки модельного эксперимента использовали ДНК, деградированную с помощью ДНКазы I в конечной концентрации 4 мЕд/мкл при инкубации в течение 3 минут при 37°С помощью термоциклера GeneAmp PCR System 9700 (AppliedBiosystems, США). Конечная концентрация ДНК в растворе составляла 0,04, 0,2 и 1 нг/мкл. Полученные образцы мужской и женской ДНК считали материалом для иллюстрации работы способа с деградированной ДНК.
Осуществляли постановку дуплексной ПЦР. Состав ПЦР-смеси на одну пробу:
10-кратный ПЦР буфер с EVAGreen– 2,5 мкл; 25 мМ MgCl2 – 2 мкл; 2,5 мМ смесь dNTP – 2,5 мкл; праймеры Amel_212-218_F/Amel_212-218_R (1 о.е.) – по 0,5 мкл; праймеры Amel_80-83_F/Amel_80-83_R (1 о.е.) – по 1 мкл; ДНК-Taq-полимераза (5 Ед/мкл) – 0,4 мкл, деионизованная вода – 12,1 мкл. Итого 22,5 мкл реакционной смеси и 2,5 мкл ДНК на 1 пробу. Конечный объем реакционной смеси равен 25 мкл.
ПЦР-реакцию проводили с соблюдением следующих температурных режимов на амплификатореi Q5 (BioRad, США):
- денатурация при 95°С – 180 с (1 цикл);
- затем 35 циклов амплификации, включающих: денатурацию при 95°С – 15 с, отжиг при 59°С – 20 с., синтез при 72°С – 30 с.;
Капиллярный гель-электрофорез полученных в ходе дуплексной ПЦР ампликонов проводили в ходе на приборе ABI PRISM 3130xl (AppliedBiosystems, США) в соответствии с руководством пользователя. Идентификацию длин амплифицированных фрагментов проводили на основе внутреннего стандарта длины (GeneScan-500 [ROX] SizeStandard).
Для загрузки образцов в прибор готовили смесь Hi-Di™ формамида и размерного стандарта GeneScan-500 [ROX] SizeStandard в следующем соотношении: 10 мклHi-Di™ формамида и 0,4 мкл размерного стандарта в расчете на один образец. Затем добавляли 1 мкл исследуемого образца.
В процессе электрофореза информация о сканировании геля лазером и детекции флуоресценции автоматически передается на управляющий компьютер и обрабатывается программой Run 3130 DataCollection v.3.0. Обработка результатов и идентификация аллелей проходит автоматически с помощью программы GeneMapper® ID 3.2.
Результаты, полученные в ходе фрагментного анализа для недеградированной ДНК (Фиг. 1А) подтверждают женский профиль ДНК, об этом свидетельствует наличие продуктов, соответствующих Х-хромосоме длиной 80 п.н. и 218 п.н. Значения «Height», выражаемые в RFU, полученные для данных пиков соответствуют низкой степени деградации ядерной ДНК.
Полученные результаты для недеградированной ДНК также подтверждают мужской профиль ДНК (Фиг. 1Б), об этом свидетельствует наличие пиков, соответствующих Х-хромосоме (продукты длиной 80 н.п. и 218 н.п.) и Y-хромосоме (продукты длиной 83 н.п. и 224 н.п.). Значения «Height», выражаемые в RFU, полученные для данных пиков соответствуют низкой степени деградации ядерной ДНК.
Несоответствие длин продуктов амплификации длинных фрагментов ДНК теоретическим связано с особенностями биоинформационной обработки данных программным обеспечением. Специфичность продуктов была подтверждена в ходе секвенирования.
Результаты, полученные для деградированной ДНК(Фиг. 1В, 1Г, 1Д) свидетельствуют о её женском профиле во всех трех случаях (интерпретация результатов аналогично вышеописанной для современной ДНК). Наблюдается разная степень деградации генетического материала: так, для начальной концентрации ДНК 0,04 нг/мкл (Фиг. 1В) наблюдается выпадение длинного фрагмента локуса амелогенина (212 н.п.). Для одних и тех же фрагментов среди трех концентраций ДНК ожидаемо отмечается разное значение высоты целевого пика.
На основании полученных данных (относительно результатов, полученных для недеградированной ДНК (Фиг 1А) можно сделать вывод о том, что деградированная ДНК в начальной концентрации 0,04 нг/мкл не рекомендуется, а ДНК в начальной концентрации 0,2 нг/мкл (Фиг 1Г) и 1 нг/мкл (Фиг 1Д) в меньшей мере рекомендуется к использованию в дальнейших исследованиях ввиду высокой вероятности непрохождения ПЦР с использованием праймеров на фрагменты генов длиной более 80 н.п.
Результаты, полученные для деградированной ДНК (Фиг 1Е, 1Ж, 1З) свидетельствуют о её мужском профиле в двух случаях из трех (интерпретация результатов аналогично вышеописанной для современной ДНК). Так, определение пола и степени деградации не представляется возможным в случае начальной концентрации ДНК 0,04 нг/мкл (Фиг 1Е) в связи с выпадением обоих локусов гена амелогенина и на Х-, и на Y-хромосомах. Наблюдается средний уровень деградации генетического материала: так, для начальной концентрации ДНК 0,2 нг/мкл (Фиг 1Ж) наблюдается выпадение обеих аллелей длинного фрагмента (212 и 218 н.п.), а для 0,1 нг/мкл (Фиг 1З) наблюдается выпадение длинного фрагмента локуса амелогенина (218н.п.), соответствующего У-хромосоме.
На основании полученных данных (относительно результатов, полученных для недеградированной ДНК (Фиг 1Б)) можно сделать вывод о том, что препарат деградированной ДНК в начальной концентрации 0,04 нг/мкл не содержит генетического материала, пригодного для генетического анализа. ДНК в начальной концентрации 0,2 нг/мкл в меньшей мере, а в начальной концентрации 1 нг/мкл в большей мере рекомендуется к использованию в дальнейших исследованиях ввиду высокой вероятности непрохождения ПЦР с использованием праймеров на фрагменты генов длиной более 80-83н.п.
Пример № 2
С целью иллюстрации способа определения наличия ингибиторов ПЦР на первом этапе осуществляли выделение ДНК из костных остатков B. taurus путем фенол-органической экстракции с предварительным этапом декальцинации и дифференциального лизиса[9].Для дополнительной очистки и концентрирования препарата ДНК использовали устройство «AmiconUltra-4, ultracel30k» (Merck, Германия).
Концентрацию и чистоту полученного препарата ДНК оценивали спектрофотометрически по соотношению коэффициентов поглощения при длинах волн 260 и 280 нм с помощью спектрофотометра ND-1000 (NanoDropTechnologies, США).
Препараты ДНК нормировали до конечной концентрации 0,25 нг/мкл и 1 нг/мкл с помощью стерильной деионизованной воды.
Для создания модели ингибирования ПЦР использовали фульвокислоты (ФК), полученных по методу Форсита из горизонта С чернозема южного [10].
Осуществляли постановку ПЦР. Состав ПЦР-смеси на одну пробу:
10-кратный ПЦР буфер с EVAGreen– 2,5 мкл; 25 мМ MgCl2 – 2 мкл; 2,5 мМ смесь dNTP – 2,5 мкл; праймеры CSN3_351_F/CSN3_351_R (1 о.е.) – по 1 мкл; ДНК-Taq-полимераза (5 Ед/мкл) – 0,5 мкл, деионизованная вода – 12мкл; ДНК B. taurus – 1 мкл. Итого 22,5 мкл реакционной смеси и 2,5 мкл ФК на 1 пробу; в случае положительного контроля вместо ФК добавляли 1 мкл деионизованной воды. Конечный объем реакционной смеси равен 25 мкл. Конечная концентрация ФК в ПЦР-смеси составляла 20 и 50 нг/мкл.
ПЦР-реакцию проводили с соблюдением следующих температурных режимов на амплификаторе iQ5 (BioRad, США):
- денатурация при 95°С – 180 с (1 цикл);
- затем 35 циклов амплификации, включающих: денатурацию при 95°С – 15 с., отжиг при 59°С – 20 с., синтез при 72°С – 30 с.;
Капиллярный гель-электрофорез полученных в ходе ПЦР ампликонов проводили в ходе на приборе ABI PRISM 3130xl (AppliedBiosystems, США) в соответствии с руководством пользователя. Идентификацию длин амплифицированных фрагментов проводят на основе внутреннего стандарта длины (GeneScan-500 [ROX] SizeStandard).
Для загрузки образцов в прибор готовили смесь Hi-Di™ формамида и размерного стандарта GeneScan-500 [ROX] SizeStandard в следующем соотношении: 10 мклHi-Di™ формамида и 0,4 мкл размерного стандарта в расчете на один образец. Затем добавляли 1 мкл исследуемого образца.
В процессе электрофореза информация о сканировании геля лазером и детекции флуоресценции автоматически передается на управляющий компьютер и обрабатывается программой Run 3130 DataCollection v.3.0. Обработка результатов и идентификация аллелей проходит автоматически с помощью программы GeneMapper® ID 3.2.
Приведены результаты фрагментного анализа, полученные с использованием двух начальных концентрации ДНКB. taurus: 0,25 нг/мкл (Фиг 2А, 2Б, 2В) и 1 нг/мкл (Фиг 2Г, 2Д, 2Е).
Говоря о результатах, полученных для ДНК в начальной концентрации 0,25 нг/мкл в случае ФК в конечной концентрации 0,2 нг/мкл (Фиг 2Б) отмечается наличие ингибиторов ПЦР. Относительно контрольного образца (Фиг. 2А) (вместо ФК в ПЦР смесь добавляли дистиллированную воду) наблюдается снижение значения RFU на 24%. Такой образец ДНК может быть к использованию в дальнейших исследованиях, однако требует применения дополнительных методик по снижению ингибирующего действия вещества (в данном случае ФК) на ПЦР. В случае ФК в конечной концентрации 50 нг/мкл (Фиг 2В) также отмечается наличие ингибиторов ПЦР Относительно контрольного образца наблюдается полное ингибирование энзиматической амплификации (снижение значения RFU на 100%). Такой образец ДНК не может быть использован в дальнейших исследованиях, без применения дополнительных методик по снижению ингибирующего действия вещества на ПЦР.
В случае результатов, полученных для ДНК в начальной концентрации 1 нг/мкл в случае ФК в конечной концентрации 0,2 нг/мкл (Фиг 2Д) отмечается наличие ингибиторов ПЦР. Относительно контрольного образца (Фиг 2Г) наблюдается снижение значения RFU на 62%. Такой образец ДНК может быть к использованию в дальнейших исследованиях, однако требует применения дополнительных методик по снижению ингибирующего действия вещества на ПЦР. В случае ФК в конечной концентрации 50 нг/мкл (Фиг 2Е) также отмечается наличие ингибиторов ПЦР Относительно контрольного образца наблюдается полное ингибирование энзиматической амплификации (снижение значения RFU на 100%). Такой образец ДНК не может быть использован в дальнейших исследованиях, без применения дополнительных методик по снижению ингибирующего действия вещества на ПЦР.
Пример № 3
С целью иллюстрации способа определения половой принадлежности, степени деградации ядерной ДНК и наличия ингибиторов ПЦР осуществляли выделение ДНК человека из клеток буккального эпителия, полученного от двух лиц (мужского и женского пола), путем фенол-органической экстракции. Для дополнительной очистки и концентрирования препаратов ДНК использовали устройство «AmiconUltra-4, ultracel30k» (Merck, Германия). Аналогичным образом проводили выделение ДНК из костных остатков B. taurus.
Концентрацию и чистоту полученных препаратов ДНК оценивали спектрофотометрически, регистрируя ультрафиолетовые спектры с помощью спектрофотометра ND-1000 (NanoDropTechnologies, США).
Препараты ДНК человека нормировали до конечной концентрации 0,2 нг/мкл, а ДНК B. taurus до 0,25 нг/мкл(использовали в качестве ВПК) с помощью стерильной деионизованной воды. Полученные образцы мужской и женской ДНК считали материалом для иллюстрации работы способа с недеградированной ДНК.
Для постановки модельного эксперимента использовали ДНК, деградированную с помощью ДНКазы I в конечной концентрации 4 мЕд/мкл при инкубации в течение 3 минут при 37°С помощью термоциклера GeneAmp PCR System 9700 (AppliedBiosystems, США). Конечная концентрация ДНК в растворе составляла 0,2 нг/мкл. Полученные образцы мужской и женской ДНК считали материалом для иллюстрации работы способа с деградированной ДНК.
Осуществляли постановку мультиплексной ПЦР. Состав ПЦР-смеси на одну пробу:
10-кратный ПЦР буфер с EVAGreen– 2,5 мкл; 25 мМ MgCl2 – 2 мкл; 2,5 мМ смесь dNTP – 2,5 мкл; праймеры Amel_212-218_F/Amel_212-218_R (1 о.е.) – по 0,5 мкл; праймеры Amel_80-83_F/Amel_80-83_R (1 о.е.) – по 1 мкл; праймеры SRY_93_F/SRY_93_R (1 о.е.) – по 1 мкл; праймеры CSN3_351_F/CSN3_351_R (1 о.е.) – по 2 мкл; ДНК-Taq-полимераза (5 Ед/мкл) – 0,5 мкл; деионизованная вода – 5 мкл; ДНК B. taurus – 1 мкл. Итого 22,5 мкл реакционной смеси и 2,5 мкл ДНК человека на 1 пробу. Конечный объем реакционной смеси равен 25 мкл.
ПЦР-реакцию проводили с соблюдением следующих температурных режимов на амплификаторе iQ5 (BioRad, США):
- денатурация при 95°С – 180 с (1 цикл);
- затем 35 циклов амплификации, включающих: денатурацию при 95°С – 15 с., отжиг при 59°С – 20 с., синтез при 72°С – 30 с.;
Капиллярный гель-электрофорез полученных в ходе мультиплексной ПЦР ампликонов проводили в ходе на приборе ABI PRISM 3130xl (AppliedBiosystems, США) в соответствии с руководством пользователя. Идентификацию длин амплифицированных фрагментов проводят на основе внутреннего стандарта длины (GeneScan-500 [ROX] SizeStandard).
Для загрузки образцов в прибор готовили смесь Hi-Di™ формамида и размерного стандарта GeneScan-500 [ROX] SizeStandard в следующем соотношении: 10 мклHi-Di™ формамида и 0,4 мкл размерного стандарта в расчете на один образец. Затем добавляли 1 мкл исследуемого образца.
В процессе электрофореза информация о сканировании геля лазером и детекции флуоресценции автоматически передается на управляющий компьютер и обрабатывается программой Run 3130 DataCollection v.3.0. Обработка результатов и идентификация аллелей проходит автоматически с помощью программы GeneMapper® ID 3.2.
Результаты, полученные в ходе фрагментного анализа для недеградированной ДНК (Фиг. 3А) подтверждают женский профиль ДНК, об этом свидетельствует наличие продуктов, соответствующих Х-хромосоме длиной 80 п.н. и 217 п.н., а также отсутствие продукта длиной 93 п.н., соответствующего Y-хромосоме. Значения «Height», выражаемые в RFU, полученные для данных пиков соответствуют низкой степени деградации ядерной ДНК. Ингибиторы ПЦР в исследуемом препарате ДНК отсутствуют или находятся на низком уровне, о чем свидетельствуют высокие значения RFU для фрагмента длиной 351 н.п., который представляет собой ВПК.
На основании полученных данных можно сделать вывод о высокой степени пригодности данного ДНК-препарата для дальнейших исследований.
Полученные результаты для недеградированной ДНК также подтверждают мужской профиль ДНК (Фиг. 3Б), об этом свидетельствует наличие пиков, соответствующих Х-хромосоме (продукты длиной 80 н.п. и 217 н.п.) и Y-хромосоме (продукты длиной 83 н.п., 224 н.п, 94 н.п.). Значения «Height», выражаемые в RFU, полученные для данных пиков соответствуют низкой степени деградации ядерной ДНК. Ингибиторы ПЦР в исследуемом препарате ДНК отсутствуют или находятся на низком уровне, о чем свидетельствуют высокие значения RFU для фрагмента длиной 350 н.п., который представляет собой ВПК.
На основании полученных данных можно сделать вывод о высокой степени пригодности данного ДНК-препарата для дальнейших исследований.
Результаты, полученные для деградированной ДНК (Фиг. 3В), свидетельствуют о её женском профиле (продукты длиной 80 н.п. и 212 н.п. – Х-хромосома). Ингибиторы ПЦР в исследуемом препарате ДНК отсутствуют или находятся на низком уровне, о чем свидетельствуют высокие значения RFU для фрагмента длиной 350 н.п., который представляет собой ВПК.
Исходя из значений RFU для полученных фрагментов относительно контрольного образца (с недеградированной ДНК (Фиг 3А), можно сделать вывод о средней степени пригодности данного ДНК-препарата для дальнейших исследований.
Результаты, полученные для деградированной ДНК (Фиг 3Г), свидетельствуют о её мужском профиле (продукты длиной 80 н.п. и 212 н.п. – Х-хромосома; 83 н.п. -Y-хромосома). Ингибиторы ПЦР в исследуемом препарате ДНК отсутствуют или находятся на низком уровне, о чем свидетельствуют высокие значения RFU для фрагмента длиной 350 н.п., который представляет собой ВПК.
Исходя из значений RFU для полученных фрагментов относительно контрольного образца (с недеградированной ДНК (Фиг 3Б)), можно сделать вывод о средней степени пригодности данного ДНК-препарата для дальнейших исследований, учитывая полное выпадение фрагмента гена SRY и длинного фрагмента гена AmelY на Y-хромосоме.
Пример № 4
С целью иллюстрации способа определения половой принадлежности, степени деградации ядерной и митохондриальной ДНК и наличия ингибиторов ПЦР осуществляли выделение ДНК человека из клеток буккального эпителия, полученного от двух лиц (мужского и женского пола), путем фенол-органической экстракции. Для дополнительной очистки и концентрирования препаратов ДНК использовали устройство «AmiconUltra-4, ultracel30k» (Merck, Германия). Аналогичным образом проводили выделение ДНК из костных остатков B. taurus.
Концентрацию и чистоту полученных препаратов ДНК проводили спектрофотометрически, регистрируя ультрафиолетовые спектры с помощью спектрофотометра ND-1000 (NanoDropTechnologies, США).
Препараты ДНК человека нормировали до конечной концентрации 0,2 нг/мкл, а ДНК B. taurus до 0,25 нг/мкл (использовали в качестве ВПК) с помощью стерильной деионизованной воды. Полученные образцы мужской и женской ДНК считали материалом для иллюстрации работы способа с недеградированной ДНК.
Для постановки модельного эксперимента использовали ДНК, деградированную с помощью ДНКазы I в конечной концентрации 4 мЕд/мкл при инкубации в течение 3 минут при 37 оС помощью термоциклера GeneAmp PCR System 9700 (AppliedBiosystems, США). Конечная концентрация ДНК в растворе составляла 0,2 нг/мкл. Полученные образцы мужской и женской ДНК считали материалом для иллюстрации работы способа с деградированной ДНК.
Осуществляли постановку мультиплексной ПЦР. Состав ПЦР-смеси на одну пробу:
10-кратный ПЦР буфер с EVAGreen– 2,5 мкл; 25 мМ MgCl2 – 2 мкл; 2,5 мМ смесь dNTP – 2,5 мкл; праймеры Amel_212-218_F/Amel_212-218_R (1 о.е.) – по 0,5 мкл; праймеры Amel_80-83_F/Amel_80-83_R (1 о.е.) – по 1 мкл; праймеры SRY_93_F/SRY_93_R (1 о.е.) – по 1 мкл; праймеры CSN3_351_F/CSN3_351_R (1 о.е.) – по 2 мкл; праймеры ND1_235_F/ND1_235_R (1 о.е.) – по 0,15 мкл; HV1_137_F/HV1_137_R (1 о.е.) – по 0,35 мкл; ДНК-Taq-полимераза (5 Ед/мкл) – 0,5 мкл; деионизованная вода – 4 мкл; ДНК B. taurus – 1 мкл. Итого 22,5 мкл реакционной смеси и 2,5 мкл ДНК человека на 1 пробу. Конечный объем реакционной смеси равен 25 мкл.
ПЦР-реакцию проводили с соблюдением следующих температурных режимов на амплификаторе iQ5 (BioRad, США):
- денатурация при 95°С – 180 с (1 цикл);
- затем 35 циклов амплификации, включающих: денатурацию при 95°С – 15 с., отжиг при 59°С – 20 с., синтез при 72°С – 30 с.;
Капиллярный гель-электрофорез полученных в ходе мультиплексной ПЦР ампликонов проводили в ходе на приборе ABI PRISM 3130xl (AppliedBiosystems, США) в соответствии с руководством пользователя. Идентификацию длин амплифицированных фрагментов проводят на основе внутреннего стандарта длины (GeneScan-500 [ROX] SizeStandard).
Для загрузки образцов в прибор готовили смесь Hi-Di™ формамида и размерного стандарта GeneScan-500 [ROX] SizeStandard в следующем соотношении: 10 мклHi-Di™ формамида и 0,4 мкл размерного стандарта в расчете на один образец. Затем добавляли 1 мкл исследуемого образца.
В процессе электрофореза информация о сканировании геля лазером и детекции флуоресценции автоматически передается на управляющий компьютер и обрабатывается программой Run 3130 DataCollection v.3.0. Обработка результатов и идентификация аллелей проходит автоматически с помощью программы GeneMapper® ID 3.2.
Результаты, полученные в ходе фрагментного анализа для недеградированной ДНК (Фиг. 4А) подтверждают женский профиль ДНК, об этом свидетельствует наличие продуктов, соответствующих Х-хромосоме длиной 80 п.н. и 217 п.н., а также отсутствие продукта длиной 93 п.н., соответствующего Y-хромосоме. Значения «Height», выражаемые в RFU, полученные для данных пиков соответствуют низкой степени деградации ядерной ДНК. Значения «Height», выражаемые в RFU, полученные для фрагментов длиной 135 п.н. и 236 п.н. (фрагменты митохондриальной ДНК) соответствуют низкой степени деградации митохондриальной ДНК. Ингибиторы ПЦР в исследуемом препарате ДНК отсутствуют или находятся на низком уровне, о чем свидетельствуют высокие значения RFU для фрагмента длиной 351 н.п., который представляет собой ВПК.
На основании полученных данных можно сделать вывод о высокой степени пригодности данного ДНК-препарата для дальнейших исследований.
Полученные результаты для недеградированной ДНК также подтверждают мужской профиль ДНК (Фиг. 4Б), об этом свидетельствует наличие пиков, соответствующих Х-хромосоме (продукты длиной 80 н.п. и 217 н.п.) и Y-хромосоме (продукты длиной 83 н.п., 224 н.п, 94 н.п.). Значения «Height», выражаемые в RFU, полученные для данных пиков соответствуют низкой степени деградации ядерной ДНК. Значения «Height», выражаемые в RFU, полученные для фрагментов длиной 135 п.н. и 235 п.н. (фрагменты митохондриальной ДНК) соответствуют низкой степени деградации митохондриальной ДНК. Ингибиторы ПЦР в исследуемом препарате ДНК отсутствуют или находятся на низком уровне, о чем свидетельствуют высокие значения RFU для фрагмента длиной 350 н.п., который представляет собой ВПК.
На основании полученных данных можно сделать вывод о высокой степени пригодности данного ДНК-препарата для дальнейших исследований.
Результаты, полученные для деградированной ДНК (Фиг. 4В), свидетельствуют о её женском профиле (продукты длиной 80 н.п. и 212 н.п. – Х-хромосома). Значения «Height», выражаемые в RFU, полученные для данных пиков соответствуют средней степени деградации ядерной ДНК. Значения «Height», выражаемые в RFU, полученные для фрагментов длиной 135 п.н. и 235 п.н. (фрагменты митохондриальной ДНК) соответствуют низкой степени деградации митохондриальной ДНК. Ингибиторы ПЦР в исследуемом препарате ДНК отсутствуют или находятся на низком уровне, о чем свидетельствуют высокие значения RFU для фрагмента длиной 350 н.п., который представляет собой ВПК.
Исходя из значений RFU для полученных фрагментов относительно контрольного образца (с недеградированной ДНК (Фиг 4А), можно сделать вывод о средней степени пригодности данного ДНК-препарата для дальнейших исследований ядерной ДНК, но высокой степени пригодности для анализа митохондриальной ДНК.
Результаты, полученные для деградированной ДНК (Фиг 4Г), свидетельствуют о её мужском профиле (продукт длиной 212 н.п. – Х-хромосома; 83 н.п., 93 н.п. -Y-хромосома). Значения «Height», выражаемые в RFU, полученные для данных пиков соответствуют средней степени деградации ядерной ДНК. Значения «Height», выражаемые в RFU, полученные для фрагментов длиной 135 п.н. и 234 п.н. (фрагменты митохондриальной ДНК) соответствуют низкой степени деградации митохондриальной ДНК. Ингибиторы ПЦР в исследуемом препарате ДНК отсутствуют или находятся на низком уровне, о чем свидетельствуют высокие значения RFU для фрагмента длиной 350 н.п., который представляет собой ВПК.
Исходя из значений RFU для полученных фрагментов относительно контрольного образца (с недеградированной ДНК (Фиг 4Б)), можно сделать вывод о средней степени пригодности данного ДНК-препарата для дальнейших исследований ядерной ДНК, учитывая полное выпадение короткого фрагмента гена AmelX и длинного фрагмента гена AmelY на Y-хромосоме. Отмечается высокой уровень пригодности для анализа митохондриальной ДНК.
Список использованных источников:
1. Корниенко И.В., Харламов С.Г. Методы исследования ДНК человека. Ростов-на-Дону: ЮФУ, 2012. – 216 с.
2. [URL: https://www.syntol.ru/catalog/nabory-dlya-opredeleniya-kontsentratsii-vydelennoy-dnk/nabor-dlya-ptsr-v-realnom-vremeni-real-quant-h3.html ]
3. [ URL: https://www.syntol.ru/catalog/nabory-dlya-opredeleniya-kontsentratsii-vydelennoy-dnk/a-detekt.html ]
4. [URL: https://evrogen.ru/products/evaluation/quantum ]
5. [ URL: https://www.qiagen-hid.ru/produkty/reagenty-dlya-real-taym-ptsr/ ]
6. Sullivan K. M. et al. A rapid and quantitative DNA sex test: fluorescence-based PCR analysis of XY homologous gene amelogenin //Biotechniques. – 1993. – Т. 15. – №. 4. – С. 637-641.
7. Haas-Rochholz H., Weiler G. Additional primer sets for an amelogenin gene PCR-based DNA-sex test //International journal of legal medicine. – 1997. – V. 110. – P. 312-315.
8. Ünal H., Kopuzlu S. The relationships between κ-casein (CSN3) gene polymorphism and some performance traits in Simmental cattle //Archives animal breeding. – 2022. – V. 65. – №. 1. – P. 129-134.
9. Корниенко И. В. и др. Y-гаплогруппы костных останков из курганных погребений хазарского времени на территории юга России //Генетика. – 2021. – Т. 57. – №. 4. – С. 464-477.
10. Влияние фульвокислот из различных почв юга России на эффективность полимеразной цепной реакции / В. С. Флоринская, И. В. Корниенко, О. Ю. Арамова [и др.] // Биотехнология. – 2023. – Т. 39, № 1. – С. 102-112.
--->
<?xml version="1.0" encoding="ISO-8859-1"?>
<!DOCTYPE ST26SequenceListing SYSTEM "ST26SequenceListing_V1_3.dtd"
PUBLIC "-//WIPO//DTD Sequence Listing 1.3//EN">
<ST26SequenceListing productionDate="2024-06-20"
softwareVersion="2.3.0" softwareName="WIPO Sequence"
fileName="Primers.xml" dtdVersion="V1_3">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2023129448/10(065504)</ApplicationNumberText>
<FilingDate>2023-11-14</FilingDate>
</ApplicationIdentification>
<ApplicantName languageCode="ru">Игорь Валериевич
Корниенко</ApplicantName>
<ApplicantNameLatin>Igor Valerievich Kornienko</ApplicantNameLatin>
<InventionTitle languageCode="ru">Способ установления половой
принадлежности, оценки степени деградации ядерной и митохондриальной
ДНК и наличия ингибиторов ПЦР с помощью фрагментного
анализа</InventionTitle>
<SequenceTotalQuantity>12</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q25">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccctttgaagtggtaccagagca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q26">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aggcttgaggccaaccatcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q27">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccctttgaagtggtaccagagca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q28">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcatgcctaatattttcagggaata</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q29">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>catcgctgttgaatacgctt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q30">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gaactctggcacctttcaat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q31">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tacattactgccagccacca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q32">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgatagttgaaggttgattgctgt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q33">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcctgccatcatgacccttg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q34">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcaggggagagtgcgtcata</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q22">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bos taurus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atttatggccattccaccaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q24">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bos taurus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>attagcccatttcgccttct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---



Изобретение относится к области генетики, молекулярной биологии, судебной медицины и криминалистики и касается инструмента для установления половой принадлежности, оценки степени деградации ядерной и митохондриальной ДНК и наличия ингибиторов ПЦР. По результатам фрагментного анализа, исходя из известных ожидаемых длин, специфичных для 4 в случае женской ДНК или 7 в случае мужской ДНК фрагментов, исследуемой ДНК человека и 1 внутреннего положительного контроля (ВПК), по наличию/отсутствию пиков alleledrop-out, их высоте и площади можно первично оценить половую принадлежность ДНК исследуемых образцов по фрагментам генов AmelX/AmelY и SRY, содержащих различной степени деградированный генетический материал, а именно ядерная ДНК – по двум фрагментам гена AmelX/AmelY и митохондриальная ДНК – по фрагментунекодирующего региона HV1 и гена ND1 и ингибиторы ПЦР - по фрагменту гена CSN3 B. taurus, представляющему собой ВПК. В результате за одну реакцию известно три параметра для конкретного препарата ДНК. Учитывая результаты, полученные с использованием описываемого способа, на последующих этапах работы исследователь получает возможность избежать неоправданной траты реактивов и времени. Техническим результатом заявляемого способа является установление половой принадлежности биологического образца, степени деградации ДНК, наличия в нем ингибиторов ПЦР при проведении мультиплексной ПЦР «в одной пробирке» и фрагментного анализа, что позволяет проводить оценку ДНК данного биологического материала для последующего исследования. 3 н. и 4 з.п. ф-лы, 4 ил., 2 табл., 4 пр.
1. Способ установления половой принадлежности, оценки степени деградации ядерной и митохондриальной ДНК и наличия ингибиторов ПЦР с помощью фрагментного анализа, который включает на первом этапе проведение дуплексной полимеразной цепной реакции с использованием ПЦР-смеси, включающей:
10-кратный ПЦР буфер – 2,5 мкл:
2,5 мМ смесь dNTP – 2,5 мкл;
25 мМ MgCl2 – 1,5-2,5 мкл;
ДНК-Taq-полимеразу 5 Ед/мкл – 0,3-0,7 мкл;
пару праймеров Amel_212-218_F/Amel_212-218_R – по 0,1-1 о.е./мкл каждого праймера;
пару праймеров Amel_80-83_F/Amel_80-83_R – по 0,5-2 о.е./мкл каждого праймера;
пару праймеров SRY_93_F/SRY_93_R – по 0,1-1,5 о.е/мкл каждого праймера;
пару праймеров ND1_235_F/ND1_235_R – по 0,05-1 о.е./мкл каждого праймера;
пару праймеров HV1_137_F/HV1_137_R – по 0,1-1 о.е./мкл каждого праймера;
пару праймеров CSN3_351_F/CSN3_351_R – по 0,5-2 о.е./мкл каждого праймера;
ДНК быка – 1 мкл в начальной концентрации 0,25-1 нг/мкл;
дистиллированную воду – до конечного объема реакционной смеси 25 мкл;
на втором этапе осуществляют анализ полученных ампликонов на ДНК-анализаторе методом капиллярного гель-электрофореза – фрагментный анализ.
2. Способ по п.1, где для контроля пола индивида используются праймеры Amel_212-218_F/Amel_212-218_R, Amel_80-83_F/Amel_80-83_R и SRY_93_F/SRY_93_R.
3. Способ по п.1, где для определения наличия ингибиторов ПЦР используют праймеры CSN3_351_F/CSN3_351_R.
4. Способ по п.1, где для оценки степени деградации митохондриальной ДНК человека используют праймеры ND1_235_F/ND1_235_R и HV1_137_F/HV1_137_R.
5. Способ по п.1, где для оценки степени деградации ядерной ДНК человека используют праймеры Amel_212-218_F/Amel_212-218_R, Amel_80-83_F/Amel_80-83_R.
6. Способ для определения наличия ингибиторов ПЦР, который включает на первом этапе проведение полимеразной цепной реакции с использованием ПЦР-смеси, содержащей ДНК данного организма в начальной концентрации 0,25-1 нг/мкл, включающей одну специфичную пару праймеров на фрагмент гена CSN3 домашнего крупного рогатого скота Bos taurus:
10-кратный ПЦР буфер – 2,5 мкл:
2,5 мМ смесь dNTP – 2,5 мкл;
25 мМ MgCl2 – 1,5-2,5 мкл;
ДНК-Taq-полимеразу 5 Ед/мкл – 0,3-0,7 мкл;
пару праймеров CSN3_351_F/CSN3_351_R – по 0,5-2 о.е./мкл каждого праймера;
ДНК быка – 1 мкл в начальной концентрации 0,25-1 нг/мкл;
дистиллированную воду – до конечного объема реакционной смеси 25 мкл;
на втором этапе осуществляют анализ полученных ампликонов на ДНК-анализаторе методом капиллярного гель-электрофореза – фрагментный анализ.
7. Способ для оценки степени деградации митохондриальной ДНК человека, который включает на первом этапе проведение дуплексной полимеразной цепной реакции с использованием ПЦР-смеси, включающей:
10-кратный ПЦР буфер – 2,5 мкл:
2,5 мМ смесь dNTP – 2,5 мкл;
25 мМ MgCl2 – 1,5-2,5 мкл;
ДНК-Taq-полимеразу 5 Ед/мкл – 0,3-0,7 мкл;
пару праймеров ND1_235_F/ND1_235_R – по 0,05-1 о.е./мкл каждого праймера;
пару праймеров HV1_137_F/HV1_137_R – по 0,1-1 о.е./мкл каждого праймера;
исследуемый образец ДНК – 2,5 мкл в начальной концентрации 0,1-1 нг/мкл;
дистиллированную воду – до конечного объема реакционной смеси 25 мкл;
на втором этапе осуществляют анализ полученных ампликонов на ДНК-анализаторе методом капиллярного гель-электрофореза – фрагментный анализ.
| Способ получения молекулярных однонуклеотидных маркеров для идентификации неизвестного индивида методом мультиплексной амплификации для работы с образцами деградированной ДНК, набор для получения молекулярных маркеров | 2021 |
|
RU2800083C2 |
| Способ получения молекулярных STR-маркеров Х-хромосомы для идентификации неизвестного индивида и определения биологического родства для работы с образцами малых количеств ДНК и набор олигонуклеотидов для его осуществления | 2021 |
|
RU2800084C2 |
| CN 110055337 A, 26.07.2019 | |||
| Набор STR-маркеров Y-хромосомы для определения этно-территориального происхождения индивида по образцу его ДНК | 2021 |
|
RU2804433C2 |
Авторы
Даты
2024-09-16—Публикация
2023-11-14—Подача