Изобретение относится к области молекулярной биологии и прогностических тестов и используется для для идентификации неизвестного индивида. Изобретение представляет собой набор синтетических олигонуклеотидных праймеров для проведения мультиплексной полимеразной цепной реакции (ПЦР). Изобретение дает возможность определить генотип по однонуклеотидным ДНК маркерам для для идентификации неизвестного индивида из образцов геномной ДНК человека, в том числе в случае, когда ДНК в исследуемом образце находится в деградированном виде.
Уровень техники
В настоящее время ДНК-анализ является наиболее доказательным методом анализа биологического материала при производстве судебно-медицинской идентификационной экспертизы. ДНК-анализ позволяет исследовать специфичные для каждого человека участки генома, и получить, таким образом, уникальный генетический "паспорт" или генетически профиль индивида. Индивидуализирующие признаки, определяемые на уровне ДНК, характеризуются почти абсолютной устойчивостью, то есть сохраняются в организме человека неизменными на продолжении всей жизни. В последние десятилетия для целей генетической идентификации личности широко применяется различные маркерные системы, локализованных как на аутосомах, так и на нерекомбинирующем участке Y-хромосомы и в митохондриальной ДНК.
Первые маркеры, которые использовали для идентификации личности были VNTR-маркеры или микросателлитные маркеры (variable number of tandem repeats) - высокополиморфные участки ДНК, представленные в геноме короткими повторяющимися последовательностями. На ранних этапах внедрения методов ДНК-идентификации в судебную практику для получения индивидуального профиля ДНК человека (ДНК-фингерпринт) использовали достаточно трудоемкую технологию, которая включала расщепление геномной ДНК специальными ферментами на короткие фрагменты, которые разделяли электрофоретически по размеру и гибридизовали с радиоактивно-мечеными пробами, соответствующими отдельным VNTR-маркерам [1, 2].
Однако, благодаря быстрым развитиям методов молекулярной биологии, в частности появлению метода полимеразной цепной реакции и ее применению в анализе ДНК, а также разработку методов прямого определения последовательности ДНК (секвенирования ДНК), ДНК-типирование стало широко использоваться в практике судебных экспертиз.
В настоящее время стандартным методом в области ДНК-идентификации является определение STR-профилей индивида с использованием метода капиллярного электрофореза. Однако, несмотря на большой прогресс в технологиях STR-анализа, есть ряд существенных проблем, которые зачастую существенно усложняют проведение генетических экспертиз путем STR-профилирования [3]. В частности, традиционный капиллярный электрофорез, широко используемый при определении STR-маркеров, основан на определении размера фрагмента ДНК. Таким образом, аллели одинаковой длины, но различной последовательности, не могут быть дискриминированы, а значит обычный фрагментный анализ STR-маркеров не может выявить точечные мутации в STR-локусах, и разрешать, например, сложные случаи отцовства [4]. Также при использовании STR-профилирования оказывается довольно затруднительно анализировать ДНК смеси от разных индивидов, что достаточно часто встречается в судебной практике [5].
В настоящее время разработаны и выпускаются наборы ДНК-маркеров на основе другого типа маркеров - SNP-маркеров, которые используются для ДНК-идентификации неизвестного индивида и установления биологического родства. При анализе SNP-маркеров отсутствуют методологические проблемы, характерные для STR-маркеров. Во-первых, очень короткие продукты амплификации, используемые для профилирования SNP, позволяют успешно применять их при анализе деградированной ДНК. Кроме того, при анализе SNP-маркеров практически исключены артефакты, обусловленные сдвигом матрицы (stutter) при амплификации STR-маркеров, и которые усложняют интерпретации профиля STR. Также, скорость мутирования SNP-маркеров существенно ниже, по сравнению с STR, что делает их более стабильными и более пригодными для анализа сложных случаев родства [3]. Секвенирование одиночных нуклеотидных полиморфизмов (SNP) вокруг и внутри STR также помогает существенно повысить эффективность анализа при определении родства [6]. Кроме того, большое количество SNP в геноме человека позволяет выбрать геномные варианты, наименее подверженные постмортальным модификациям ДНК, например, те, в которых не участвует цитозин (чтобы исключать постмортальные замены цитозина на урацил), или те, которые расположены в близком контакте с нуклеосомами, а значит защищены нуклеосомными белками и таким образом менее подвержены постмортальным химическим модификациям. В геномах людей существуют миллионы одно нуклеотидных полиморфизмов в отличие от сотен STR-маркеров. Это позволяет более гибко выбирать используемые для анализа SNP.
Значительным технологическим новшеством последних лет стало появление технологий секвенирования нового поколения масштабного параллельного секвенирования (NGS). Существенным преимуществом данного типа анализа является то, что с их применением стало возможным проведение анализа одновременно всех типов маркеров, основанных на SNP, STR и митохондриальной ДНК (мтДНК). Основная проблема которая может быть успешно решена на основе технологии NGS это способность различать аллели, имеющие аналогичную длину, что является необходимой предпосылкой для идентификации смесей образцов [4].
Преимущества использования SNP-маркеров в сочетании с методом массового параллельного секвенирования (NGS):
- возможность анализа деградированной ДНК (за счет уменьшения длинны ампликона)
- одновременный анализ сотен образцов
- возможность значительного увеличения точности системы за счет увеличения количества маркеров, анализируемых в одной пробирке (в мультиплексе)
На текущий день уже разработан ряд панелей SNP, достаточных для достоверного определения совпадения ДНК-профилей на уровне, сравнимом с анализом на основе STR-маркеров. Например, была разработана панель IISNP из 45 однонуклеотидных замен с помощью которой возможна ДНК-идентификация с точностью до 10-15. Расширенная версия панели, включающая 86 SNP, уменьшает вероятность ошибки до 10-30. Эта панель применяется в коммерческих системах MiSeq FGx™ Forensic Genomics System и HID-Ion AmpliSeq™ Identity Panel [7], которая использует технологии масштабного параллельного секвенирования для генотипирования. Кроме того, была предложена панель SNPforID из 52 маркеров, позволяющая получить вероятность случайного совпадения SNP-профилей 10-18 и используемая в коммерческой системе HID-Ion AmpliSeq™ Identity Panel [8]. В настоящее время SNP-маркеры для ДНК-идентификации (IISNP) используются в качестве аналога аутосомных STR маркеров для сравнения эталонных образцов с неизвестными образцами [7].
Имеющиеся в настоящее время разработки зарубежных наборов SNP-маркеров не учитывают специфики популяционной структуры населения России (большое число этнических групп, специфические для различных этнических групп полиморфизмы, которые не учитываются в иностранных разработках). В настоящее время на территории РФ нет разработки наборов для идентификации неизвестного индивида на основе SNP-маркеров, которые могли быть использованы в качестве более эффективного аналога применяемых в настоящее время зарубежных коммерческих наборов Для успешного применения SNP-маркеров для задач отечественной криминалистики и судебной медицины необходима разработка методик генотипирования и наборов SNP-маркеров, которая позволит максимально охватить генетическое разнообразие по этим локусам в российских популяциях. Кроме этого, немаловажным недостатком существующих импортных наборов для ДНК-идентификации является их значительная дороговизна, обусловленная необходимостью использования дорогостоящих расходных материалов (специфических ДНК-полимераз, растворов для ПЦР, буферных растворов). Для устранения указанных недостатков необходимым условием является повышение информативности предлагаемого набора и его экономичности, а также адаптации для специфики используемых в криминалистических экспертизах образцов биологического материала, где ДНК сильно деградирована или содержится в малом количествах.
Для решения поставленной задачи авторами предложен набор из 95 пар синтетических олигонуклеотидных праймеров, комплементарных участкам локусов SNP-маркеров для выявления генотипов, позволяющих проводить идентификации личности в российских популяциях.
Настоящее изобретение раскрывает разработанные синтетические оригинальные олигонуклеотидные праймеры, позволяющие амплифицировать локусы, содержащие целевые молекулярно-генетические маркеры для последующего секвенирования с помощью метода «массового параллельного секвенирования». Набор для генотипирования включает композицию, образованную 95 парами синтетическими оригинальными олигонуклеотидными праймерами для локусов в геноме человека, включающих SNP-маркеры, позволяющую проводить мультиплексную ПЦР (нуклеотидная последовательность праймеров SEQ ID NO: 1- SEQ ID NO 190, приведена в таблице 1). Синтетические олигонуклеотидные праймеры подобраны таким образцом, чтобы ПЦР-продукт имел минимальную длину, меньше 120 п. н., что позволяет успешно анализировать сильно деградированные образцы ДНК.
Преимущество данного набора состоит в том, что авторами изобретения создана система олигонуклеотидов, позволяющая одновременно анализировать множество образцов, учитывающая популяционный полиморфизм, встречающийся в российских популяциях, простое обнаружение с использованием высокопроизводительных технологий (NGS-секвенирование), а также предназначенная для использования на малых количествах биологического материала низкого качества, в том числе, криминалистических образцов, в который ДНК находится в сильно деградированном состоянии.
Выбор набора маркеров
Для эффективного использования биаллельных SNP маркеров для идентификации индивида необходимо увеличить их количество для достижения дискриминационного потенциала сравнимого с уровнем дискриминации STR маркеров. Число используемых однонуклеотидных вариантов должно быть в 3-1 раза больше чем STR маркеров[9] Однонуклеотидные маркеры для идентификации личности должны быть высоко гетерозиготны и обладать низкой частотой вариаций в разных популяциях. Основываясь на этих требованиях были разработаны первые SNP-панели, которые включали 40-50 SNP, которые обеспечивают вероятность идентификации, сравнимую со стандартными STR-панелями. Например, набор 45 несцепленных автономных SNP, который обеспечивает вероятность идентификации в пределах от 10-16 до 10-19 [10, 11] или панель "SNPforID" содержащая 52 SNP [12, 8]. Такие панели маркеров применимы к лицам европейского происхождения, но обладают немного меньшей дискриминационной способностью у неевропейцев. Эти панели SNP маркеров используются в коммерческих наборах для идентификации. Сейчас на рынке имеется две лидирующие панели для идентификации личности на основе SNP маркеров. "HID-Ion AmpliSeq Identity Panel" [13] включает 48 SNP из панели "SNPforID" и 43 маркера из панели К. Кидда. Совокупная вероятность совпадения генотипов в этой панели варьирует для разных популяций в пределах от 1 × 10-31 до 1 × 10-33. "ForenSeq DNA Signature Prep Kit" для приборов компании "Illumina" [14] содержит 94 SNP для идентификации из тех же панелей.
Критерии для выбора олигонуклеотидных последовательностей праймеров были следующими:
- Разработанная пара олигонуклеотидов должна обеспечивать специфическую амплификацию конкретного участка генома человека, содержащего выбранный ДНК маркер
- На последовательности олигонуклеотида не должно содержаться известных полиморфизмов, согласно базе данных gnomAD
- Температуры отжига разработанных олигонуклеотидов должны варьироваться от 57 до 58 градусов Цельсия
- Все разработанные пары олигонуклеотидов должны быть совместимы друг с другом, то есть не образовывать вторичных структур, чтобы избежать ингибирования амплификации некоторых геномных фрагментов при проведении мультиплексной ПЦР.
Так как разрабатываемая панель специально предназначена для работы с деградированной ДНК важно было минимизировать размер ампликона, содержащего маркер. Поэтому основным критерием для отбора SNP-маркера в панель был размер амплифицированного фрагмента, содержащего выбранный маркер, который не должен был превышать 120 пар оснований.
В результате были отобраны 95 одно нуклеотидных маркеров (Таблица 1).






Авторы изобретения провели анализ данных "массового параллельного секвенирования" на приборах Illumina, полученных для фрагментных библиотек, приготовленных на основе ПЦР-продуктов мультиплексной амплификации. Данный анализ подтвердил эффективность разработанной системы олигонуклеотидов для определения генотипов по всем выбранным 95 позициям для SNP маркеров. Кроме того, было проведено полногеномное секвенирование около 20 образцов ДНК различных индивидов и сравнение полученных профилей с данными секвенирования мультиплексных ПЦР-продуктов, содержащих набор разработанных маркеров. В результате анализа было выявлено 100%-ное соответствие генотипов полногеномного секвенирования с генотипами маркеров в ПЦР продуктах, полученными с помощью данного набора олигонуклеотидов на тех же образцах ДНК.
Осуществление способа
Пример генотипирования биологического образца.
В примере возможность идентификации неизвестного индивида показана на образцах геномной ДНК человека путем проведения мультиплексного ПЦР-анализа с последующим "массовым параллельным секвенированием" полученных ПЦР-продуктов на технологической платформе Illumina.
Для проведения ДНК-идентификации используется образец ДНК в количестве не менее 100 пкг, выделенная из любого биоматериала человека.
Получение ДНК
ДНК может быть получена из любой ткани человека, содержащей клеточные ядра, например, лейкоцитов, волосяных фолликулов, клеток буккального эпителия и др. Пригодная для проведения ПЦР ДНК может быть выделена из образа при помощи любого ранее широко применяемого метода выделения ДНК должен быть в равной степени эффективным. В частности, есть несколько готовых наборов для выделения ДНК из различных тканей человека (например, представленных фирмой Qiagen)
Идентификация неизвестного индивида с помощью панели олигонуклеотидных праймеров для 95 SPN-маркеров включает в себя четыре этапа:
- Мультиплексная ПЦР геномной ДНК с использованием смеси специфических олигонуклеотидных праймеров;
- Приготовление фрагментных библиотек из полученных ПЦР-продуктов для секвенирования на платформе Illumina;
- Проведение широкомасштабного параллельного секвенирования полученных библиотек;
- Анализ данных секвенирования и интерпретация результатов.
Амплификация участков генома, содержащих исследуемые маркеры
Амплификация участков генома, содержащих исследуемые маркеры, может быть осуществлена с помощью наборов Multiplex PCR kit компании «Qiagen», на приборах GeneAmp PCR System 9700 Thermal Cycler компании «Applied Biosystems».
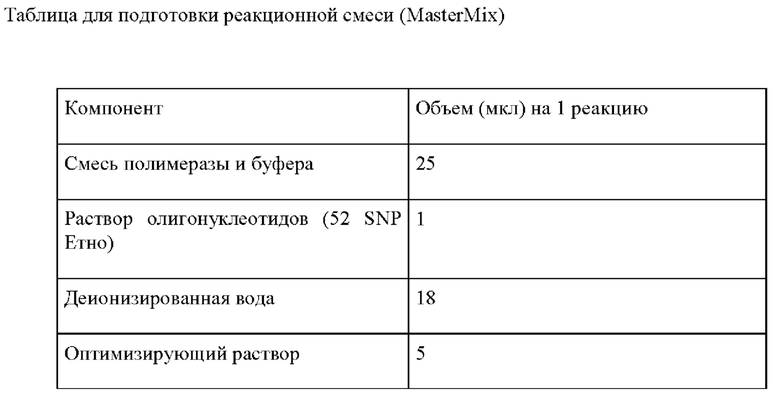
Состав реакционной смеси для мультиплексной ПЦР:
- Раствор олигонуклеотидов для мультиплексной ПЦР (в конечной концентрации от 1 нМ до 0,5 мкМ
- Смесь полимеразы и буфера
- Деионизированная вода
- Оптимизирующий раствор
- исследуемая ДНК
Оптимальное рекомендуемое количество анализируемой ДНК от 100 пг до 10 нг.При необходимости образец ДНК следует развести в буфере ЕВ.
Также необходимо проводить параллельно контрольную реакцию (отрицательный контроль), где вместо ДНК добавляется деионизированная вода.
Амплификацию следует проводить в пробирках, стрипах, или планшетах предназначенных для ПЦР.
В отдельной стерильной пробирке смешать из расчета на одну реакцию все необходимые компоненты, согласно расчетной таблице (пробирка с раствором MasterMix).
После приготовления, смесь для амплификации помещают в амплификатор и выставляется программа:
95°С - 15 минут
94°С - 30 секунд
57°С - 90 секунд 35 циклов
72°С - 90 секунд
72°С - 10 минут
4°С ∞.
Наличие продуктов амплификации исследуемой ДНК и отсутствие продуктов амплификации в отрицательном контроле проверяют с помощью электрофореза в агарозном геле. Продукты амплификации очищают от остаточных праймеров с помощью соответствующих мини-колонок (например, Qiagen MinElute system) в соответствии с инструкцией к используемому набору реагентов. Полученные в результате мультиплексной амплификации очищенные ПЦР продукты, содержащие маркеры для идентификации неизвестного индивида используются в качестве матрицы для приготовления фрагментных геномных библиотек для секвенирования.
Приготовление фрагментных библиотек
Предварительно необходимо провести измерение концентрации очищенных ПЦР-фрагментов, полученных на предыдущем этапе мультиплексной амплификации на приборах NanoDrop или Qubit (ThermoFisher) в соответствии с инструкцией к прибору. Для приготовления фрагментных библиотек оптимально использовать около 300 нг ДНК очищенных ПЦР-продуктов мультиплексной амплификации. Приготовление фрагментных геномных библиотек проводят с использованием наборов реагентов Illumina®. TruSeq® DNA PCR-Free library prep kit в соответствии с инструкцией к набору. Для секвенирования нескольких образцов фрагментных библиотек одновременно необходимо использовать индексированные адаптеры (например, Dual index adaptors Illumina). Для каждого образца библиотеки используется отдельный индексируемый адаптер, который необходимо записать в таблицу для дальнейшего учета при анализе результатов секвенирования нескольких образцов фрагментных библиотек. Для приготовления фрагментных библиотек рекомендуется/возможно использовать 1/2 от объема всех реагентов, входящих в набор Illumina®. TruSeq® DNA PCR-Free library prep kit, прописанного в инструкции к набору.
Этапы 1-3 являются рекомендованными, но не обязательными.
1 Очистка фрагментных библиотек: необходимо нанести весь объем элюата в одну лунку 2% агарозного геля с красителем SYBR Gold (например, с использованием готового 2%-ного E-gel (Invitrogen)). Провести электрофорез с использованием маркера молекулярных весов 50 bp Ladder в течение 10 минут (в случае использования системы e-gel (Invitrigen)).
2 с использованием стерильного одноразового скальпеля вырезать кусочки геля, с фрагментами библиотеки от 220 п. н. и больше. Светящиеся фрагменты длиной меньше 150 п. н. соответствуют димерам адаптеров и не должны быть использованы для дальнейшего анализа.
3 Провести очистку вырезанных фрагментов ДНК с использованием набора реагентов MinElute gele extraction kit (Qiagen). Эллюировать в 20-22 мкл буфера ЕВ (Qiagen).
Приготовленные описанным выше способом фрагментные библиотеки можно напрямую использовать для секвенирования на технологической платформе Illumina, предварительно проведя оценку качества полученных фрагментных библиотек.
Качественную и количественную оценку фрагментных библиотек можно проводить стандартными методами, рекомендованными фирмой производителем платформ для секвенирования Illumina. Для оптимизации рабочего процесса рекомендуем использовать следующие этапы: измерить концентрации фрагментных библиотек на приборе Qubit с использованием набора реагентов HS. Для дальнейшего секвенирования рекомендуется использовать фрагментные библиотеки с концентрацией не менее 2 нМ/мкл.
Перед секвенированием необходимо провести мультиплексирование библиотек в эквимолярных количествах (объединение библиотек для нескольких образцов). Не допускается объединение в один пул библиотек с одинаковыми индексами.
Приготовленные фрагментные библиотеки секвенируют методом параллельного широкомасштабного секвенирования на технологической платформе Illumina в парноконцевом режиме с длинной прочтения не менее 75+75 п.о. Рекомендуемый прибор секвенатор MiSeq (Illumina).
Анализ результатов секвенирования
Анализ результатов секвенирования проводят следующим образом:
Картировать полученные прочтения на референсный геном человека (GRCh37) с помощью программы bwamem или аналогичной.
Определить генотипы по исследуемым маркерам с помощью программ GATK, freebayes или аналогичных.
Литературные источники:
1. Devesse L., Ballard D., Davenport L. et al. Concordance of the ForenSeq system and characterisation of sequence-specific autosomal STR alleles across two major population groups // Forensic Sci. Int. Genet. 2017. V. 34. P. 57-61. doi 10.1016/j.fsigen.2017.10.012
2. Phillips C, Gettings K.B., King J.L. et al. "the devil's in the detail": release of an expanded, enhanced and dynamically revised forensic STR sequence guide // Forensic Sci. Int. Genet. 2018. V. 34. P. 162 169. doi 10.1016/j.fsigen.2018.02.017
3. Gettings K.B., Borsuk L.A., Ballard D. et al. STRSeq: a catalog of sequence diversity at human identification short tandem repeat loci // Forensic Sci. Int. Genet. 2017. V. 31. P. 111-117. doi 10.1016/j.fsigen.2017.08.017
4. van der Gaag K.J., de Leeuw R.H., Hoogenboom J. et al. Massively parallel sequencing of short tandem repeats-population data and mixture analysis results for the PowerSeq system // Forensic Sci. Int. Genet. 2016. V. 24. P. 86-96. doi 10.1016/j.fsigen.2016.05.016
5. Phillips C, Fang R., Ballard D. et al. Evaluation of the Genplex SNP typing system and a 49plex forensic marker panel // Forensic Science International-Genetics. 2007. V. 1(2). P. 180-185. doi 10.1016/j.fsigen.2007.02.007
6. Tozzo P., Gabbin A., Politi C. et al. Combined Statistical Analyses of Forensic Evidence in Sexual Assault: A Case Report and Brief Review of the Literature // J. Forensic Sci. 2020. V. 65. P.1767-1773. doi 10.1111/1556-4029.14487
7. Fei Guo, Yishu Zhou, He Song, Jinling Zhao, Hongying Shen, Bin Zhao, Feng Liu, Xianhua Jiang, Next generation sequencing of SNPs using the HID-Ion AmpliSeq™ Identity Panel on the Ion Torrent PGM™ platform, Forensic Science International: Genetics, Volume 25, 2016, Pages 73-84, ISSN 1872-4973, https://doi.org/10.1016/j.fsigen.2016.07.021.
8. Musgrave-Brown E, Ballard D, Balogh K, Bender K, Berger B, Bogus M et al. Forensic validation of the SNPforlD 52-plex assay. Forensic Science International-Genetics. 2007 Jun;l(2): 186-90. https://doi.org/10.1016/j.fsigen.2007.01.004
9. Krawczak M. 1999. Informativity assessment for biallelic single nucleotide polymorphisms. Electrophoresis. 20, 1676-1681. Berglund E.C., Kiialainen A., Syvanen A.-C. 2011. Next-generation sequencing technologies and applications for human genetic history and forensics. Investigative Genetics. 2, 23.
10. Pakstis A.J., Speed W.C., Fang R., Hyland F.C.L., Furtado M.R., Kidd J.R., Kidd K.K. 2010. SNPs for a universal individual identification panel. Hum Genet. 127, 315-324.
11. Kidd K.K. 2011. Population Genetics of SNPs for Forensic Purposes (Updated). NIJ Final Report. 103 p.
12. Sanchez J.J., Phillips C., Borsting C., Balogh K., Bogus M., Fondevila M., Harrison C.D., Musgrave-Brown E.,Salas A., Syndercombe-Court D., Schneider P.M., Carracedo A., Morling N. 2006. A multiplex assay with 52 single nucleotide polymorphisms for human identification. Electrophoresis. 27, 1713 1724.
13. Churchill J.D., Chang J., Ge J., Rajagopalan S.C. Wootton C.W. 2015. Blind study evaluation illustrates utility of the Ion PGM system for use in human identity DNA typing. Croat. Med. J. 56, 218 229., Life technologies. 2014. HID-Ion AmpliSeq Identity Panel Data Sheet. 2 P.
14. Illumina. 2014. ForenSeq DNA Signature Prep Kit Data Sheet. 4 P
Список нуклеотидных последовательностей
SEQ IID1
GAACTGCCTGGTGTGGACT
SEQ IID2
CCACACCTGAACCAGACACT
SEQ IID3
AGTGACCAGGACTGTACGTGT
SEQ IID4
CGGTACCCAAGAAGACTCTG
SEQ IID5
AGGAGTTCCCACCAGTTTCT
SEQ IID6
CAGGATGCAAACTCTTGGAG
SEQ IID7
TACCCTCTCTGCTTCTGGAG
SEQ IID8
TGAGTCTGGGAAAAACAACA
SEQ IID9
CATTTTCATCAAATTTCCATTC
SEQ IID10
TGTGTGAGCTATGACACTCCTT
SEQ IID11
GAATCCTGGTCAACAACCTC
SEQ IID12
CTGCAACATTCCATTATCCA
SEQ IID13
CATGCAGGCTCCATTTTTAT
SEQ IID14
AGCAGGCAGTTAGCAGAGTG
SEQ IID15
TGGTGAGGGTGGAGATTTTA
SEQ IID16
GTCTCAAGGTCGTGCCAGT
SEQ IID17
GGTGATTTTTCCCATGATGA
SEQ IID18
TGCTTTGTAAGGCAATAGAGC
SEQ IID19
TGATCAATTGTTTGTCAGAATGT
SEQ IID20
CAGCTCTGATGATGTGCAAG
SEQ IID21
CCCAGGGAGTTCCTGATAAC
SEQ IID22
AGCCTTGGAAAACACAGAAA
SEQ IID23
TGACTTCCCAAGCTGAATTT
SEQ IID24
TGAGAATTGAAAAGCCCAAT
SEQ IID25
TGTAACCCAGCAATTCCAGT
SEQ IID26
GGGGCAACTATGAATACAGC
SEQ IID27
AGCCAAGATCATGTCAGTGC
SEQ IID28
CAAGTTTGTTGGCTTCTTTTG
SEQ IID29
CCCTCACAGTTAAAGCTTTTGT
SEQ IID30
ATGACCTGGATTGATCAGAGA
SEQ IID31
CCATGATTTTCTTGTGGTGA
SEQ IID32
CATGTGCTTAGGCCACAAC
SEQ IID33
AAGGTTAGGGATTGGAATGG
SEQ IID34
CGTAGTAAATGAGAGCAAGTTT
SEQ IID35
TCTGCCAAATGTATCCTTACC
SEQ IID36
TGGTCATTGTTGACACTTCAC
SEQ IID37
TGGCAACTTCTGATAAAGGAT
SEQ IID38
TTCAGTAGTTAATATCAAGGAAGTTT
SEQ IID39
CCACACCTCCAGATGAGAGT
SEQ IID40
ATAAGGCAAGGATGAACAGG
SEQ IID41
AAGGTGCAAGGAATTAGCAG
SEQ IID42
CGGCTTTTGTCTTCTCTTCT
SEQ IID43
СTGTGTGTTTTAAAGCCAGG
SEQ IID44
ACCAGGCATTTGACCTTCTA
SEQ IID45
TTCCACGAAAGTTCTTCTCC
SEQ IID46
CATAGCTTGTGTTGGTCAGG
SEQ IID47
AAGCTTTAGAAAGGCATATCGT
SEQ IID48
TTGGGGGAGCTAAACCTAAT
SEQ IID49
GATTGACTGTTTCTCATCCTGTT
SEQ IID50
ATGGTTTTTAGGCACCATGA
SEQ IID51
TTCAGTGAATAAATGGGCTCT
SEQ IID52
CTGTGGCAAAATGAAGAATG
SEQ IID53
AGAGGGTGGGCAAAGTTATT
SEQ IID
TCTGGGGCAGATGAAGTAGT
SEQ IID54
CTCTCTCTCCTGCTCTCTGG
SEQ IID55
GAAGAATCCAGCCCTTGTC
SEQ IID56
TGCTGTGGACTGAAACTTGA
SEQ IID57
CCATCCCAGCTGAGTATTCC
SEQ IID58
GAATGAATTTGAAAAAGCATCA
SEQ IID59
CCTTTCTGTTTTGTCCATCTG
SEQ IID60
CATCAACTTTATCGCTTTTTCC
SEQ IID61
GGATGCTTGCAAACAAAGAC
SEQ IID62
TGAGCGTTTTCCTCTGTTTT
SEQ IID63
TCATGAGATTGCTGGGAGAT
SEQ IID64
AGTTGGGATAGGTGATGCAA
SEQ IID65
GTTCTTTTCTCCGGGCTAAC
SEQ IID66
CCATGAAGATGGAGTCAACA
SEQ IID67
CAACCAGAGCACAAGTGGTA
SEQ IID68
GGTTACCTGTTTTCCTTTTGTG
SEQ IID69
ATGTGCGTTTCTCCACACTT
SEQ IID70
CAGTCTCTCTGTGCCGAAG
SEQ IID71
GCCCTTACTGTGATGTAGGC
SEQ IID72
AGTGGCATTAGAAATTCCAGA
SEQ IID73
CAGTATCCCCGCAAACTAAC
SEQ IID74
GAGACTCAGTGTTCAAGGTCAC
SEQ IID75
GAGGCACACTCAGATGCAC
SEQ IID76
AACTGGGTGTTAGGGAGACA
SEQ IID77
GCGTTCTGTATAGGCACCAT
SEQ IID78
GCCTGTGGCTTTGTAGTTCT
SEQ IID79
GAAGGGCAGTGAGGAGTTC
SEQ IID80
TTGTCATTCTGATGGACTGG
SEQ IID81
AACATTGCCTCTCCTTTGTT
SEQ IID82
GGCAGCATACACTCATAGCC
SEQ IID83
GTGTGCATTGTCTCGTGTGT
SEQ IID84
CTGGGGAGTAGAGGAGACCT
SEQ IID85
GCTTCGCTTTGCTACTCTTC
SEQ IID86
ACCCAGTGTTCCCAGCTT
SEQ IID87
TGTTGACCCCGTCGTATCTA
SEQ IID88
CTATTGCCTACAAATTTCAATTACA
SEQ IID89
AATTTCCCAGCAAAAACTTCT
SEQ IID90
AGAAGGGACCTAACCTGGAG
SEQ IID91
AGTGTCACCGAATTCAACG
SEQ IID92
GGTGTGTATTCTGCGGTAGC
SEQ IID93
GAAACATGGGATGAACAAGG
SEQ IID94
CAAATAGCAATGGCTCGTCT
SEQ IID95
AGGGTTTGAGCAGTTCTGAG
SEQ IID96
GGAGGTGATTTCTGTGAGGA
SEQ IID97
AAGAACCAGAGGTTGCTGTC
SEQ IID98
TGAGATAAATCAGACCAGGATG
SEQ IID99
CCCTTCTCAGCACTTCCATA
SEQ IID100
CTATGTCAGTCTGGGTGCAA
SEQ IID102
TGGGCATCTGGAATGTACTA
SEQ IID102
SEQ IID 103
CCCACTTGTTCTTTTTCTGC
SEQ IID104
CGAAACATGCTTCAGAGGTTA
SEQ IID105
ACAGGTATCCTGGCCTCAC
SEQ IID106
TCCCATGAAACTTTTCAACTC
SEQ IID107
CACTTTTCCAGCTGTCCCTA
SEQ IID108
GCTGGAATCCCGCTTACTA
SEQ IID109
CTGAATCCAGCCATGTTGTA
SEQ IID110
GCAAGATCTTTGCCAGTGA
SEQ IID111
GACCAGCTTCTGTCTGGAAG
SEQ IID112
CACTGTCCATCACGACACC
SEQ IID113
GCAGGGTGCAGGTATGTATT
SEQ IID114
TCAACAAACGTGTGATGCTC
SEQ IID115
TTGTCAATCTTTCTACCAGAGG
SEQ IID116
CTTATGACGCCTGGATTTTC
SEQ IID117
TGGTTCAAGGTTCTGGAGTT
SEQ IID118
TTGTTCTTCTCCATCCCATT
SEQ IID119
TGGCTTCTCTTTCCCTTATG
SEQ IID120
AGGTTGCGATAGAAAACAGTG
SEQ IID121
GGGGAGGAGGGAAATACA
SEQ IID122
GCAGCTGTCCATCATCAGTA
SEQ IID123
GGTAGGACCAGAAGTGTTTCA
SEQ IID124
TTGTGTTTAAACTTGGATACCATC
SEQ IID125
GATAGCCCTGCATTCAAATC
SEQ IID126
CTCTGGTTCACAAATGAGCA
SEQ IID127
CGATGATGACTTCCCAGAG
SEQ IID128
CTTCCTCAGTAGCAGCACCT
SEQ IID129
CTTTGTGTGGCTGAGAGAGA
SEQ IID1130
ATCTGGCTCACTGGTTGTTT
SEQ IID131
ACAATGGAGCCACTGAACTG
SEQ IID132
CTTTGTGTGGCTGAGAGAGA
SEQ IID133
ATCTTCCTCCTGGAGATCAA
SEQ IID134
GCAAGTATGTTTGGCAAATG
SEQ IID135
AAGCAAAGCAAAGCCTCAT
SEQ IID136
AAGTAACACATTTCCCTCTTGC
SEQ IID137
TCAAAAAGTTGTCAGCATGG
SEQ IID138
AACCAGCAACACTCCTAATCA
SEQ IID139
TGAATGGTGTGATGTAAACG
SEQ IID140
CCACCTATGGGCTCTTCTTA
SEQ IID141
GCACGAAGGAGAAACACCT
SEQ IID142
AGCAAAAATCCCTTAGGATG
SEQ IID143
ATTCATGAGCTGGTGTCCAA
SEQ IID144
AGGGACAAAGCTGACAAGC
SEQ IID145
GTTTGCTAAGTAAGGTGAGTGG
SEQ IID146
AGGTTCGAGTTTTGGCTTTA
SEQ IID147
ACCACCTGCCTCTCATCTT
SEQ IID148
GCTGGCAGAACAGAGAGATATT
SEQ IID149
GGAGGAGTCTTAGAGGAGGTG
SEQ IID150
GACTCAGCTACTGCCAACCT
SEQ IID151
CCAAAGCTATTCTCTCTTTTGG
SEQ IID152
TGAGAAAACTTGAAAATGTTGG
SEQ IID153
GATGCAACATGAGAGAGCAG
SEQ IID154
AGGTCACGAGCTCAATTTTC
SEQ IID155
GACAGTTAAGAGAAGGCTGCTT
SEQ IID156
CCCCACTCAACACACAGAA
SEQ IID157
TTATCCTTTCCTGCTTTTCC
SEQ IID158
GAGAGACGATAGGTCTGTAAGGA
SEQ IID159
CTCCCATCAACCTCTTTTGT
SEQ IID160
ATGATAGTGGCAAAGGAGAGA
SEQ IID161
CTTTCCCACATTATGGTCCT
SEQ IID162
CCCTATGCCAAGGATATAACA
SEQ IID163
ACCCATCACACTATCCTGACA
SEQ IID164
CACCTGGCCTACAATTCAAA
SEQ IID165
GGGAGGAGACAGCTTCTTG
SEQ IID166
ACTTTAGCCACCAAAATCCA
SEQ IID176
GGTTTGTGTGTGAGTGTTTCA
SEQ IID178
CAGTCCTCCAGCTCCTTATG
SEQ IID169
TTTTTCTGAAGCCCTTTCAT
SEQ IID170
CCCATGCAGAAATGAATGTA
SEQ IID171
AATCCCCGTTCACTTAGATG
SEQ IID172
GAAATATTCAGCACATCCAA
SEQ IID173
CCATGAGACTGGGTTCACTT
SEQ IID174
AGGCTCTGAATCAGGATGAG
SEQ IID175
AATTGGGGACCTATGCTGT
SEQ IID176
CCAACAATTTCCCTAGAACC
SEQ IID177
CGAGAATACAAGCAGCAGAG
SEQ IID178
AGACCAGTCACCTGTTTTGC
SEQ IID179
CCTCTGAGATGATGAATGCTT
SEQ IID180
GTCCATGCTAGAAAAAGCTGA
SEQ IID181
CAAGTGGGGAAACTAGGTCA
SEQ IID182
CAGTGCCACTCTTCACTGAG
SEQ IID183
AAGTACACCCCAGGCTCAG
SEQ IID184
CTCGCTTGAGTTTTCTTTGG
SEQ IID185
CACTGTTTGGCATGAACTTG
SEQ IID186
CCATGCCATCGTCTGTATTA
SEQ IID187
TCTGGAATGCCAGTTCTTTT
SEQ IID188
AGAACGCCTATGAAAACCAG
SEQ IID189
CTCCCTGACCTGTGGTCTT
SEQ IID190
GGATGAAGGTTAGAGCCAGA
Изобретение относится к области молекулярной биологии, биотехнологии, судебной медицины и криминалистики, генетическим исследованиям в области идентификации человека. Представлен способ получения молекулярных однонуклеотидных маркеров для идентификации неизвестного индивида методом мультиплексной амплификации для работы с образцами деградированной ДНК; набор для получения молекулярных маркеров. Изобретение раскрывает разработанные синтетические оригинальные олигонуклеотидные праймеры, позволяющие амплифицировать локусы, содержащие целевые однонуклеотидные маркеры для последующего секвенирования с помощью метода «массового параллельного секвенирования». Набор для генотипирования включает композицию, образованную 95 парами синтетических оригинальных олигонуклеотидных праймеров для локусов в геноме человека, включающих однонуклеотидные маркеры, позволяющую проводить мультиплексную ПЦР. Последовательность праймеров приведена в SEQ ID NO: 1 - SEQ ID NO 190. Синтетические олигонуклеотидные праймеры подобраны таким образцом, чтобы ПЦР-продукт имел минимальную длину, меньше 120 п.н. Изобретение позволяет успешно анализировать деградированные образцы ДНК, идентифицировать неизвестного индивида и определить биологическое родство в российской популяции с высокой чувствительностью, специфичностью и точностью по сравнению с существующими аналогами. 2 н. и 4 з.п. ф-лы, 2 табл., 1 пр.
1. Способ получения молекулярных однонуклеотидных маркеров для идентификации неизвестного индивида методом мультиплексной амплификации для работы с образцами деградированной ДНК, характеризующийся тем, что для генотипирования используют композицию из 95 пар оригинальных синтетических олигонуклеотидных праймеров для проведения мультиплексной полимеразной цепной реакции (ПЦР) с нуклеотидными последовательностями SEQ ID NO: 1 - SEQ ID NO: 190, фланкирующих участки ДНК, содержащие однонуклеотидные вариации (SNP); ПЦР-продукты, соответствующие выбранным праймерам, представляют собой маркеры.
2. Способ по п. 1, отличающийся тем, что размер полученных ПЦР-продуктов составляет не более 120 п.н.
3. Способ по п. 1, отличающийся тем, что полученные в результате мультиплексной амплификации ПЦР-продукты, содержащие маркеры для идентификации неизвестного индивида, используются в качестве матрицы для различных методов генетического определения нуклеотидной последовательности.
4. Способ по п. 1, отличающийся тем, что определение нуклеотидной последовательности ПЦР-продуктов, содержащих маркеры для идентификации неизвестного индивида, проводят секвенированием по методу «массового параллельного секвенирования» (NGS).
5. Способ по п. 1 для идентификации неизвестного индивида, происходящего из популяций Российской Федерации.
6. Набор для получения молекулярных маркеров способом по п. 1, используемых для генотипирования индивида, используемый для детектирования присутствия молекулярных однонуклеотидных SNP-маркеров, предназначенных для идентификации неизвестного индивида, включающий оригинальные синтетические олигонуклеотидные праймеры с нуклеотидными последовательностями SEQ ID NO: 1 - SEQ ID NO 190.
| ForenSeq DNA Signature Prep Kit Data Sheet: Forensic Genomics " Illumina", 2015 | |||
| Очаг для массовой варки пищи, выпечки хлеба и кипячения воды | 1921 |
|
SU4A1 |
| Д | |||
| В | |||
| РЕБРИКОВ и др., ПЦР в реальном времени, 6-е издание, БИНОМ, лаборатория знаний, Москва, 2015, стр | |||
| Устройство для выпрямления опрокинувшихся на бок и затонувших у берега судов | 1922 |
|
SU85A1 |
| Основы полимеразной | |||
Авторы
Даты
2023-07-18—Публикация
2021-11-10—Подача