Изобретение относится к области молекулярной генетики и может быть использовано для выявления генотипов для идентификации личности и определения этнической принадлежности человека по образцу его ДНК с помощью системы микросателлитных ДНК-маркеров Y-хромосомы.
Типирование ДНК является наиболее доказательным методом анализа биологического материала при производстве судебно-медицинской идентификационной экспертизы. Молекулярно-генетический идентификационный анализ позволяет исследовать особые участки ДНК, строго специфичные для каждого индивидуума, и получить, таким образом, уникальный генетический "паспорт" человека, которое нельзя скрыть, изменить или подделать. Индивидуализирующие признаки, определяемые на уровне ДНК, характеризуются почти абсолютной устойчивостью, то есть сохраняются в организме человека неизменными всю его жизнь.
В течение последнего десятилетия для целей генетической идентификации личности широко применяется изучение различных маркерных систем, локализованных на аутосомах, нерекомбинирующем участке Y-хромосомы и в митохондриальном геноме. Преимущество нерекомбинирующих линий Y-хромосомы заключается в том, эти обладая достаточно высокой информативностью и для «общей» ДНК-идентификации, передаются строго от отца к сыну, что позволяет напрямую устанавливать родство [1, 2].
Среди мировых приоритетных направлений в области генетики человека и медицинской генетики в настоящее время на первый план выступают разработки тест-систем, направленных на создание высокотехнологичной услуги по определению генетического происхождения индивида по мужской линии; разработки новых или усовершенствования существующих генетических тест-систем, применяемых в судебной медицине для идентификации личности и определения отцовства [3, 4]
Полиморфизм, используемый в качестве основной системы генетического маркирования, применяемой в судебно-медицинской практике, возникает в результате изменения числа идентичных ДНК-повторов (STR) в пределах одного локуса. Преимущество STR для идентификации личности и оценки биологического родства заключается в возможности проведения успешного анализа даже при использовании сильно деградировавших образцов ДНК. Развитие технологий генотипирования позволяет легко автоматизировать генетический анализ микросателлитов.
ДНК-идентификация личности основывается на анализе частоты встречаемости маркеров аутосомных локусов, Y-хромосомы и мтДНК [5, 6]. Анализ Y-хромосомы и (или) мтДНК необходим в случаях, когда нужно установить родственные отношения по отцовской или материнской линиям; используется при работе с сильно деградированными образцами ДНК вследствие очень высокой копийности мтДНК; а также может быть альтернативой ДНК-идентификации по аутосомным локусам [7] Ключевым моментом в ДНК-идентификации являются вероятностные расчеты совпадения генотипов, основанные на референтных частотах аллелей в популяции, из которой происходит исследуемый ДНК-профиль.
Преимущество STR для идентификации личности и оценки биологического родства заключается в возможности проведения успешного анализа даже при использовании сильно деградировавших образцов ДНК. Ранее судебно-медицинским стандартом являлся "минимальный гаплотип" из 7 YSTR, который позднее был вытеснен "расширенным гаплотипом" из 9 локусов. На сегодняшний день созданы YSTR-наборы, используемые для различных целей, в том числе и для идентификации личности.
В настоящее время разработаны и выпускаются следующие наборы ДНК-маркеров Y-хромосомы, которые используются для ДНК-идентификации:
AmpFlSTR® Yfiler Kit (фирмы Applied Biosystems), позволяющий амплифицировать 17 STR-локусов Y-хромосомы в одной ПЦР-реакции, который состоит из реакционной ПЦР-смеси, смеси праймеров, ДНК-полимер азы, контрольной ДНК и аллельного лэддера.
PowerPlex®Y23 System (фирмы Promega) для исследования коротких тандемных повторов, позволяющий амплифицировать 23 STR-локуса Y-хромосомы в одной ПЦР-реакции, который состоит из реакционной ПЦР-смеси, смеси праймеров, ДНК-полимеразы, контрольной ДНК, аллельного лэддера и деионизованой воды.
Investigator Argus Y-12 QS Kit (фирмы Qiagen) для ПЦР-амплификации для исследования коротких тандемных повторов, позволяющий амплифицировать 12 STR-локусов Y-хромосомы в одной ПЦР-реакции, который состоит из реакционной ПЦР-смеси, смеси праймеров, ДНК-полимеразы, контрольной ДНК и аллельного лэддера.
Y-FilerPlus (фирмы Applied Biosystems) для исследования коротких тандемных повторов, позволяющий амплифицировать 25 STR-локусов в одной ПЦР реакции.
Имеющиеся в настоящее время разработки наборов STR-маркеров Y-хромосомы не учитывают специфики популяционной структуры населения России (большое число этнических групп, специфические для различных этнических групп полиморфизмы, не учтенные в иностранных разработках). Для успешного применения Y-STR в криминалистике и судебной медицине необходима разработка методик генотипирования и наборов Y-STR, которая позволит максимально охватить генетическое разнообразие по этим локусам в российских популяциях. Кроме этого, существенным недостатком существующих наборов является их значительная дороговизна. Из-за необходимости использования специальных дорогостоящих расходных материалов (специфических ДНК-полимераз и аллельных лэддеров) существенно ограничиваются возможности широкого применения существующих наборов в диагностических лабораториях. Для устранения указанных недостатков необходимым условием является повышение информативности предлагаемого набора и его экономичности.
В реестре изобретений Российской федерации фигурирует 3 сходных патента (RU 2558231, RU 2528742, RU 2740575), при этом только один из трех является дейсвующим (RU 2740575). Ближайшими аналогами патентуемого нами набора STR-маркеров Y-хромосомы являются RU 2528742 (Синтетические олигонуклеотидные праймеры и способ выявления генотипов для идентификации личности с помощью системы микросателлитных ДНК-маркеров Y-хромосомы) и набор Y-FilerPlus.
Разработанный нами набор STR-маркеров Y-хромосомы, позволяет определять популяционную принадлежность индивида мужского пола по специфичности его YSTR-гаплотипа. В результате специфичности наследования и отсутствия рекомбинации Y-хромосомы популяционная специфичность различных линий (гаплотипов и субгаплогрупп), при выбранном числе используемых STR-маркеров, позволяет достаточно точно проводить определение этно-территориального происхождения индивида мужского пола (по отцовской линии). Выбранные микросателлитные маркеры являются достаточно высокоинформативными для большинства популяционных выборок. Применение этого набора YSTR-маркеров, позволило перейти на более точный новый уровень детализации и дифференциации популяций. Уровень информативности гаплотипов по выбранным маркерам позволяет достичь значений, практически позволяющих идентифицировать различия между популяционными группами. В результате анализа полученных генотипических данных было показано, что гаплотипы, построенные с помощью генотипирования 38 STR-маркеров Y-хромосомы обладает значительной популяционной и территориальной специфичностью. Установлена очень высокая межпопуляционная дифференциация полученных YSTR-гаплотипов, что позволяет эффективно использовать отработанный набор YSTR-маркеров для целей ДНК-идентификации.
Новая техническая задача - повышение чувствительности, точности и информативности способа и создание системы генетических маркеров охватывающей генетическое разнообразие по 38 локусам в российских популяциях.
Для решения поставленной задачи предложены синтетические олигонуклеотидные праймеры (SNP ID NO 1-70), комплементарные соответствующим участкам пятнадцати микросателлитных локусов Y-хромосомы, причем используют пары праймеров, один из которых имеет флуоресцентную метку, а второй является немеченым, при этом, прямые праймеры для каждого из локусов несут на 5'-конце флуоресцентную метку: ТЕТ (4, 7, 2', 7'-тетрахлоро-6-карбоксифлуоресцеин) для DYS385, DYS390, DYS391, DYS426, DYS436 и DYS439; FAM (6-карбоксифлуоресцеин) для DYS392, DYS393, DYS437 и DYS438; и HEX (4, 7, 2', 4',5',7'-гексахлоро-6-карбоксифлуоресцеин) для DYS394, DYS389I, DYS389II DYS388 и DYS434, также, синтетические олигонуклеотидные праймеры имеют следующий нуклеотидный состав:
DYS385 (SNP ID NO: 1) F: 5'-TET-AGCATGGGTGACAGAGCTA-3'
DYS385 (SNP ID NO: 2) R: 5 '-TGGGATGCTAGGTAAAGCTG
DYS389 (SNP ID NO: 3) F: 5'-HEX-CCAACTCTCATCTGTATTATCTATG
DYS389 (SNP ID NO: 4) R: 5'-TCTTATCTCCACCCACCAGA
DYS390 (SNP ID NO: 5) F: 5'-TET-TATATTTTACACATTTTTGGGCC
DYS390 (SNP ID NO: 6) R: 5'-TGACAGTAAAATGAACACATTGC
DYS391 (SNP ID NO: 7) F: 5'-TET-CTATTCATTCAATCATACACCCA
DYS391 (SNP ID NO: 8) R: GATTCTTTGTGGTGGGTCTG
DYS392 (SNP ID NO: 9) F: 5'-FAM-TCATTAATCTAGCTTTTAAAAACAA
DYS392 (SNP ID NO: 10) R: 5'-AGACCCAGTTGATGCAATGT
DYS393 (SNP ID NO: 11) F: 5'-FAM-GTGGTCTTCTACTTGTGTCAATAC
DYS393 (SNP ID NO: 12) R: 5'-AACTCAAGTCCAAAAAATGAGG
DYS394 (SNP ID NO: 13) F: 5'-HEX-CTACTGAGTTTCTGTTATAGT
DYS394 (SNP ID NO: 14) R: 5'-ATGGCATGTAGTGAGGACA
DYS437 (SNP ID NO: 15) F: 5'-FAM-GACTATGGGCGTGAGTGCAT-3'
DYS437 (SNP ID NO: 16) R: 5'-GAGACCCTGTCATTCACAGATGA
DYS438 (SNP ID NO: 17) F: 5'-FAM-CCAAAATTAGTGGGGAATAGTTG-3'
DYS438 (SNP ID NO: 18) R: 5'-GATCACCCAGGGTCTGGAGTT-3'
DYS439 (SNP ID NO: 19) F: 5'-TET-TCGAGTTGTTATGGTTTTAGGTCT-3'
DYS439 (SNP ID NO: 20) R: 5'-GTGGCTTGGAATTCTTTTACCC-3'
DYS442 (SNP ID NO: 21) F: 5'-HEX-CCCCAAGTCCCCAAAGTGTGT-3'
DYS442 (SNP ID NO: 22) R: 5'-CACTCATTGATTGATGGGCGTTT-3'
DYS445 (SNP ID NO: 23) F: 5'-FAM-AGTTAAGAGCCCCACCTTCCTG-3'
DYS445 (SNP ID NO: 24) R: 5'-TTTTGGTGGCATAATCTCAGCTC-3'
DYS448 (SNP ID NO: 25) F: 5'-FAM-TGGGAGAGGCAAGGATCCAA-3'
DYS448 (SNP ID NO: 26) R: 5'-CCAGACCGGCCAGAAATATGAC-3'
DYS449 (SNP ID NO: 27) F: 5'-FAM-TTTTCCCTTAACTTGTGTGATTTTT-3'
DYS449 (SNP ID NO: 28) R: 5'-AGGTTGGACAACAAGAGTAAGACAG-3'
DYS456 (SNP ID NO: 29) F: 5'-FAM-GGACCTTGTGATAATGTA-3'
DYS456 (SNP ID NO: 30) R: 5'-GTTTTGGGCTGAGTTGATGGG-3'
DYS458 (SNP ID NO: 31) F: 5'-FAM-AGCAACAGGAATGAAACTCCAAT-3'
DYS458 (SNP ID NO: 32) R: 5'-GGAGGGTGGGCGTGGTGG-3'
DYS460 (SNP ID NO: 33) F: 5'-ROX-GAGGAATCTGACACCTCTGACA-3'
DYS460 (SNP ID NO: 34) R: 5'-GTCCATATCATCTATCCTCTGCCTA-3'
DYS481 (SNP ID NO: 35) F: 5'-JOE-AGGAATGTGGCTAACGCTGT-3'
DYS481 (SNP ID NO: 36) R: 5'-ACAGCTCACCAGAAGGTTGC-3'
DYS504 (SNP ID NO: 37) F: 5'-HEX-TCTACACCACTGTGCCAAGC-3'
DYS504 (SNP ID NO: 38) R: 5'-GGCAACAGAGCAACCCTCT-3'
DYS505 (SNP ID NO: 39) F: 5'-FAM-TCTGGCGAAGTAACCCAAAC-3'
DYS505 (SNP ID NO: 40) R: 5'-TCGAGTCAGTTCACCAGAAGG-3'
DYS518 (SNP ID NO: 41) F: 5'-JOE-GGCAACACAAGTGAAACTGC-3'
DYS518 (SNP ID NO: 42) R: 5'-TCAGCTCTTACCATGGGTGAT-3'
DYS525 (SNP ID NO: 43) F: 5'-HEX-ATTCACACCATTGCACTCCA-3'
DYS525 (SNP ID NO: 44) R: 5'-GAGGCGGAGCTTTTAGTGAG-3'
DYS531 (SNP ID NO: 45) F: 5'-FAM-GACCCACTGGCATTCAAATC-3'
DYS531 (SNP ID NO: 46) R: 5'-TGCTCCCTTTCTTTGTAGACG-3'
DYS533 (SNP ID NO: 47) F: 5'-JOE-CATCTAACATCTTTGTCATCTACC-3'
DYS533 (SNP ID NO: 48) R: 5'-TGATCAGTTCTTAACTCAACCAA-3'
DYS537 (SNP ID NO: 49) F: 5'-HEX-GGTCTCCAATTCCATCCAGA-3'
DYS537 (SNP ID NO: 50) R: 5'-TGGAACATGCCCATTAATCA-3'
DYS552 (SNP ID NO: 51) F: 5'-FAM-CCATAGTGCCGAGGTCAAGT-3'
DYS552 (SNP ID NO: 52) R: 5'-AACACCTG ATGCCTGGTTG- 3'
DYS570 (SNP ID NO: 53) F: 5'-ROX-GAACTGTCTACAATGGCTCACG-3'
DYS570 (SNP ID NO: 54) R: 5'-TCAGCATAGTCAAGAAACCAGACAAC-3'
DYS576 (SNP ID NO: 55) F: 5'-ROX-TGGGCTGAGGAGTTCAATC-3'
DYS576 (SNP ID NO: 56) R: 5'-GGCAGTCTCATTTCCTGGAGATG
DYS635 (SNP ID NO: 57) F: 5'-FAM-AGTGTCTCACTTCAAGCACCAAGCAC-3'
DYS635 (SNP ID NO: 58) R: 5'-ACATTTTTCCAACTGTGAATTTTGCTGC-3'
DYS643 (SNP ID NO: 59) F: 5'-FAM-AAGCCATGCCTGGTTAAACT-3'
DYS643 (SNP ID NO: 60) R: 5'-TGTAACCAAACACCACCCATT-3'
YCAII (SNP ID NO: 61) F: 5'-HEX-TGTCAAAATTTAACCCACAATCA-3'
YCAII (SNP ID NO: 62) R: 5'-GCAGTCTTTCACCATAAGGTTAGC-3'
GATA H4.1 (SNP ID NO: 63) F: 5'-HEX-ATGCTGAGGAGAATTTCCAA-3'
GATA H4.1 (SNP ID NO: 64) R: 5'-GCTATTCATCCATCTAATCTATCCATT-3'
Y-GATA-A10 (SNP ID NO: 65)
F: 5'-NED-CCTGCCATCTCTATTTATCTTGCATATA-3'
Y-GATA-A10 (SNP ID NO: 66) R: 5'-ATAAATGGAGATAGTGGGTGGATT-3'
GGAAT1B07 (SNP ID NO: 67) F: 5'-NED-CATTAACCATAGCATTTCTTTCC-3'
GGAAT1B07 (SNP ID NO: 68) R: 5'-GAAGTAGAGTGGAATGGACTGC-3'
DYF387S1 (SNP ID NO: 69) F: 5'-FAM-GCCTGGGTGACAGAGCTAGA-3'
DYF387S1 (SNP ID NO: 70) R: 5'-GCCACAGTGTGAGAAGTGTGA-3'
В способе выявления генотипов для идентификации личности с помощью системы микросателлитных ДНК-маркеров Y-хромосомы используют вышеуказанные праймеры, при этом, проводят генотипирование 38 коротких тандемных повторов, локализованных на нерекомбинирующем участке Y-хромосомы (YSTR), на генетическом ДНК-анализаторе с помощью капиллярного гель-электрофореза, с применением метода ПЦР и фрагментного анализа флуоресцентно меченых ДНК-ампликонов, используют образцы ДНК мужчин, выделенную из любого биологического материала в соответствии со стандартными методами, обеспечивающими качественную очистку ДНК от белков и примесей и предотвращающими деградацию материала в ходе выделения, при проведении капиллярного электрофореза фрагменты, синтезированные в ходе ПЦР, имеющие флуоресцентную метку ТЕТ (4, 7, 2', 7'-тетрахлоро-6-карбоксифлуоресцеин) в диапазоне длин 360-420 п.о. соответствуют аллелям 9-23 тандемных повтора в локусе DYS385, фрагменты, имеющие флуоресцентную метку ТЕТ в диапазоне длин 196-224 п.о. соответствуют аллелям 19-26 тандемных повторов в локусе DYS390, фрагменты, имеющие флуоресцентную метку ТЕТ в диапазоне длин 277-289 п.о. соответствуют аллелям 9-12 тандемных повторов в локусе DYS391, фрагменты, имеющие флуоресцентную метку ТЕТ в диапазоне длин 232-260 п.о. соответствуют аллелям 8-15 тандемных повторов в локусе DYS439, фрагменты, имеющие флуоресцентную метку FAM (6-карбоксифлуоресцеин) в диапазоне длин 243-259 п.о. соответствуют аллелям 10-15 тандемных повторов в локусе DYS392, фрагменты, имеющие флуоресцентную метку FAM в диапазоне длин 116-128 п.о. соответствуют аллелям 12-15 тандемных повторов в локусе DYS393, фрагменты, имеющие флуоресцентную метку FAM в диапазоне длин 174-190 п.о. соответствуют аллелям 6-10 тандемных повторов в локусе DYS437, фрагменты, имеющие флуоресцентную метку FAM в диапазоне длин 311-326 п.о. соответствуют аллелям 9-12 тандемных повторов в локусе DYS438, фрагменты, имеющие флуоресцентную метку FAM в диапазоне длин 311-326 п.о. соответствуют аллелям 9-12 тандемных повторов в локусе DYS438, фрагменты, имеющие флуоресцентную метку FAM в диапазоне длин 251-263 п.о. соответствуют аллелям 9-12 тандемных повторов в локусе DYS445, фрагменты, имеющие флуоресцентную метку FAM в диапазоне длин 307-343 п.о. соответствуют аллелям 18-24 тандемных повторов в локусе DYS448, фрагменты, имеющие флуоресцентную метку FAM в диапазоне длин 264-313 п.о. соответствуют аллелям 25-37 тандемных повторов в локусе DYS449, фрагменты, имеющие флуоресцентную метку FAM в диапазоне длин 141-157 п.о. соответствуют аллелям 17-17 тандемных повторов в локусе DYS456, фрагменты, имеющие флуоресцентную метку FAM в диапазоне длин 106-156 п.о. соответствуют аллелям 12-24 тандемных повторов в локусе DYS458, фрагменты, имеющие флуоресцентную метку FAM в диапазоне длин 165-181 п.о. соответствуют аллелям 11-15 тандемных повторов в локусе DYS505, фрагменты, имеющие флуоресцентную метку FAM в диапазоне длин 102-127 п.о. соответствуют аллелям 8-14 тандемных повторов в локусе DYS531, фрагменты, имеющие флуоресцентную метку FAM в диапазоне длин 236-256 п.о. соответствуют аллелям 7-12 тандемных повторов в локусе DYS552, фрагменты, имеющие флуоресцентную метку FAM в диапазоне длин 238-274 п.о. соответствуют аллелям 17-26 тандемных повторов в локусе DYS635, фрагменты, имеющие флуоресцентную метку FAM в диапазоне длин 123-158 п.о. соответствуют аллелям 7-14 тандемных повторов в локусе DYS643, фрагменты, имеющие флуоресцентную метку FAM в диапазоне длин 240-272 п.о. соответствуют аллелям 27-35 тандемных повторов в локусе DYF387, фрагменты, имеющие флуоресцентную метку HEX (4, 7, 2', 4', 5', 7'-гексахлоро-6-карбоксифлуоресцеин) в диапазоне длин 190-206 п.о. соответствуют аллелям 13-17 тандемных повторов в локусе DYS394, фрагменты, имеющие флуоресцентную метку HEX в диапазоне длин 242-262 п.о. соответствуют аллелям 7-12 тандемных повторов в локусе DYS389I, фрагменты, имеющие флуоресцентную метку HEX в диапазоне длин 360-382 п.о. соответствуют локусу DYS389II, аллели локуса DYS389II определяют вычитанием из значения длины фрагмента диапазона 360-382 значения длины фрагмента ВУ83891,получаемый диапазон 110-134 п.о. соответствует 14-20 тандемным повторам, фрагменты, имеющие флуоресцентную метку HEX в диапазоне длин 298-322 п.о. соответствуют аллелям 10-16 тандемных повторов в локусе DYS442, фрагменты, имеющие флуоресцентную метку HEX в диапазоне длин 265-296 п.о. соответствуют аллелям 12-20 тандемных повторов в локусе DYS504, фрагменты, имеющие флуоресцентную метку HEX в диапазоне длин 305-321 п.о. соответствуют аллелям 9-13 тандемных повторов в локусе DYS525, фрагменты, имеющие флуоресцентную метку HEX в диапазоне длин 155-179 п.о. соответствуют аллелям 7-13 тандемных повторов в локусе DYS537, фрагменты, имеющие флуоресцентную метку HEX в диапазоне длин 147-165 п.о. соответствуют аллелям 17-26 тандемных повторов в локусе YCAII, фрагменты, имеющие флуоресцентную метку HEX в диапазоне длин 134-152 п.о. соответствуют аллелям 9-14 тандемных повторов в локусе Y-GATA-H4.1, фрагменты, имеющие флуоресцентную метку ROX (карбокси-Х-родамин) в диапазоне длин 102-122 п.о. соответствуют аллелям 8-13 тандемных повторов в локусе DYS460, фрагменты, имеющие флуоресцентную метку ROX в диапазоне длин 242-282 п.о. соответствуют аллелям 13-23 тандемных повторов в локусе DYS570, фрагменты, имеющие флуоресцентную метку ROX в диапазоне длин 168-204 п.о. соответствуют аллелям 13-22 тандемных повторов в локусе DYS576, фрагменты, имеющие флуоресцентную метку JOE в диапазоне длин 117-157 п.о. соответствуют аллелям 189-32 тандемных повторов в локусе DYS481, фрагменты, имеющие флуоресцентную метку JOE (б-карбокси-4',5'-дихлор-2',7'-диметоксифлуоресцеин) в диапазоне длин 252-296 п.о. соответствуют аллелям 25-36 тандемных повторов в локусе DYS518, фрагменты, имеющие флуоресцентную метку JOE в диапазоне длин 200-224 п.о. соответствуют аллелям 9-15 тандемных повторов в локусе DYS533.
Предлагаемый способ заключается в генотипировании 38 STR-маркеров Y-хромосомы на генетическом ДНК-анализаторе с помощью капиллярного гель-электрофореза, с применением метода ПЦР и фрагментного анализа флуоресцентно меченых ДНК-ампликонов. Разработанная система генетических маркеров Y-хромосомы (YSTR) информативна для ДНК-идентификации в популяциях России и основана на мультиплексном генотипировании методом ПЦР и капиллярного гель-электрофореза.
Способ предназначен:
- для выявления генотипов микросателлитных (STR) локусов Y-хромосомы человека в образцах ДНК, полученных из биологического материала лиц мужского пола для генетической идентификации личности человека в медико-диагностических, судебно-медицинских и криминалистических лабораториях;
- для установления этнической принадлежности человека по структуре его гаплотипу YSTR;
- для установления родственных связей, в том числе при исследовании случаев спорного отцовства;
- для научных исследований в области популяциогнной генетики человека и медицинской генетики.
Разработанный способ заключается в использовании специально подобранных высокоспецифичных флуоресцентно-меченых олигонуклеотидных праймеров для проведения полимеразной цепной реакции (ПЦР) по амплификации микросателлитных маркеров Y-хромосомы человека.
Предлагаемый способ позволяет получить комплексный генотип исследуемого образца ДНК человека по 38 микросателлитным маркерам нерекомбинирующей части Y-хромосомы: DYS19, DYS385a, DYS385b, DYS389I, DYS389II, DYS390, DYS391, DYS392, DYS393, DYS437, DYS438, DYS439, DYS442, DYS445, DYS448, DYS449, DYS456, DYS458, DYS460, DYS481, DYS504, DYS505, DYS518, DYS525, DYS533, DYS537, DYS552, DYS570, DYS576, DYS635, DYS 643, YCAIIa, YCAIIb, GATA-H4.1, Y-GATA-A10, GGAAT1B07, DYF387a, DYF387b.
Генотипы представлены в виде размеров длин фрагментов соответствующих ампликонов в нуклеотидах. Именно такой широкий набор YSTR является очень информативным для проведения популяционно-генетических и криминалистических исследований. Оценка информативности выбранных маркеров по доступным литературным данным и генетическим базам данных показала высокий потенциал данного набора микросателлитных маркеров нерекомбинирующей части Y-хромосомы для генетической дифференциации популяций и индивидов и привязки человека к конкретной этнической группе по специфичности его гаплотипа в пределах различных гаплогрупп Y-хромосомы.
Способ осуществляют следующим образом
Для работы используют образцы ДНК, выделенной из любого биологического материала человека в соответствии со стандартными методами, обеспечивающими качественную очистку ДНК от белков и примесей и предотвращающими деградацию материала в ходе выделения. Концентрация рабочего раствора тотальной недеградированной ДНК для проведения ПЦР должна быть не менее 2 нанограмм на микролитр.
Генотипирование микросателлитных ДНК-маркеров проводят в несколько этапов, включающих проведение подготовки образцов ДНК, полимеразной цепной реакции (ПЦР), пробоподготовки продуктов амплификации, капиллярный гель-электрофорез на автоматических ДНК-анализаторах, компьютерную программную обработку первичных данных и, собственно, получение итоговых генотипов исследуемых образцов.
Мультиплексное генотипирование включает в себя несколько последовательных шагов:
- получение ПЦР-продуктов, содержащих последовательности с изучаемыми YSTR у исследуемого образца;
- объединение в общей смеси ПЦР-продуктов всех исследуемых маркеров;
- очистка смеси ампликонов от минерального масла (если ПЦР проводилась с добавлением минерального масла в амплификаторах без нагревающейся крышки);
- приготовление смеси продуктов ПЦР с матричным стандартом длины и формамидом;
- денатурация продукта реакции в формамиде;
- капиллярный электрофорез на автоматическом анализаторе;
- программная обработка первичных данных;
- получение итоговых генотипов исследуемых образцов.
Для амплификации выбранных YSTR-маркеров проводят полимеразную цепную реакцию. Для эффективного разделения всех продуктов в соответствии с их физическим размером на капиллярном гель-электрофорезе праймеры подобраны соответствующим образом. Предлагаемые праймеры, выбраны из множества вариантов для наиболее оптимального сочетания длин амплифицированных продуктов, разделения флуоресцентных меток, эффективной дифференциации близких по размеру аллелей разных локусов и сочетания красителей со стандартными наборами реагентов для оптической калибровки автоматических ДНК-анализаторов.
Каждый из используемых красителей имеет свою длину волны максимума флюоресценции (хотя спектры эмиссии перекрываются). Затем компьютерная программа анализирует интенсивность флюоресценции и распознает пики продуктов ПЦР, исходя из того, на какой длине волны имело место максимальное увеличение флюоресценции по сравнению с фоновым уровнем. Результаты в итоге представлены в виде разноцветных пиков (каждый из цветов соответствует определенному флуоресцентному красителю) -электрофоре граммы, с обозначенным размера продуктов, который устанавливается по стандарту длины, добавляемому при пробоподготовке в каждый образец.
Праймеры на локусах DYS385, DYS389, YCAII и DYF387 подобраны таким образом, что одновременно амплифицируется, соответственно, по два микросателлитных повтора DYS385a, DYS385b, DYS389II, дающий фрагмент большей длины (353-385 п.н.), и DYS389I, которому соответствуют фрагменты длиной 239-263 п.н. (Cooper et al., 1996). Число повторов в локусе DYS389II определяют, вычитая из длины большего фрагмента длину меньшего.
ПЦР проводят в пробирках или плашках объемом 0,2 или 0,5 мл. Объем реакционной смеси может варьировать 10 до 50 мкл. Рекомендуемый объем 15 мкл. Реакционная смесь для ПЦР, общим объемом 15 мкл включает в себя по 0,5-1,5 мкл специфических праймеров с концентрацией 1 о.е./мл; 0,2 мМ концентрацию каждого из четырех dNTP (обычно 1 мкл 8 мМ раствора, в том числе 2 мМ каждого dNTP); от 10 до 100 нг исследуемого образца геномной ДНК; буфер для Taq-полимеразы и 0,25-1,0 ед. Taq-ДНК-полимеразы. Реакцию проводят в специализированных термоциклерах (амплификаторах).
Подбор условий проведения полимеразной цепной реакции с использованием набора флуоресцентно меченых праймеров включал оценку количества и качества выхода продуктов амплификации всех пятнадцати STR-локусов, наличие неспецифической амплификации, наличие различных аберраций при проведении капиллярного электрофореза, различия в интенсивности высоты детектируемых пиков продуктов, количество праймеров, не включившихся в реакцию. Проведено сравнение различных буферов, различной концентрации ионов Mg++, концентрации смеси праймеров, концентрации dNTP и общего объема ПЦР-смеси. В результате были подобраны оптимальные условия для проведения ПЦР.
Рекомендуется использовать современные амплификаторы для 96-луночных тонкостенных плашек: Applied Biosystems, Forster Sity, США; Biometra, ФРГ; I-Cycler, BioRad, США. Схема полимеразной цепной реакции состоит из первичной денатурации ДНК-матрицы (4 минут при 94-95°С), затем 40 циклов, состоящих из денатурации (94°С), отжига праймеров при специфической для каждого полиморфизма температуре (обычно от 58°С до 62°С), элонгации цепи (при 72°С); реакцию завершает финальная элонгация цепи (5-10 минут при 72°С).
Программа для амплификации локусов DYS19, DYS389I, DYS389II, DYS390, DYS391, DYS392, DYS437, DYS442, DYS445, DYS448, DYS449, DYS481, DYS505, DYS518, DYS525, DYS533, DYS537, DYS552, GATA-H4.1:
Начальная денатурация: 94°С - 4 мин.
40 циклов: 94°С - 30 сек
58°С - 30 сек
72°С - 45 сек
Заключительная элонгация: 72°С - 4 мин.
Программа для амплификации локусов DYS385a, DYS385b, DYS393, DYS438, DYS439, DYS456, DYS458, DYS460, DYS481, DYS570, DYS576, DYS635, DYS 643, YCAIIa, YCAIIb, Y-GATA-A10, GGAAT1B07, DYF387a, DYF387b:
Начальная денатурация: 94°C - 4 мин.
40 циклов: 94°C - 30 сек
60°C - 30 сек
72°C - 45 сек
Заключительная элонгация: 72°С - 4 мин.
Программа для амплификации локусов DYS437, DYS438:
Начальная денатурация: 94°С - 4 мин.
40 циклов: 94°С - 30 сек
62°С - 30 сек
72°С - 45 сек
Заключительная элонгация: 72°С - 4 мин.
Сравнительное тестирование различных ДНК-полимераз российского производства, рекомендованных производителями для проведения ПЦР. Сравнивались четыре различные ДНК-полимеразы. Самые лучшие результаты, по итогам всех испытаний получены с применением Hot Start Taq ДНК полимеразы компании СибЭнзим (Новосибирск).
Полученные продукты ПЦР отдельных локусов объединяют в денатурирующем буфере. Для этого, в отдельных пробирках (или плашках) смешивают равное количество продуктов ПЦР всех исследуемых YSTR-маркеров (амликонов). Рекомендуемый объем каждого маркера 5 мкл.
Все ПЦР-продукты разводят деионизованной водой до концентрации 10 пМ/мкл (10 кмкМ раствор). Исходная концентрация зависит от качества и концентрации исходной ДНК-матрицы, оптимизации ПЦР, применяемой полимер азы и расходных материалов. Относительные пропорции смешиваемых продуктов в условиях работы конкретной лаборатории могут потребовать коррекции, проводящейся опытным путем.
Реакция денатурации ПЦР-продуктов в формамиде ставится в общем объеме 10 мкл. В пробирки или плашки для автоматического анализатора переносят по 2 мкл смеси ПЦР-продуктов, 0,5 мкл маркера молекулярной массы (Gene-Scan-500 ROX size standart), 9 мкл Hi-Di формамида. Размерный стандарт представляет собой смесь искусственно синтезированных олигонуклеотидных фрагментов длиной от 35 до 500 пар оснований (35, 50, 75, 100, 139, 150, 160, 200, 250, 300, 340, 350, 400, 450, 490 и 500) с химически привитой флюорисцентной группой, находящуюся в растворе, при облучении флюорисцирует в спектре красного цвета. Полученную смесь денатурируют 5 мин. при 95°С, затем сразу охлаждают, либо во льду, либо при 4°С (5 минут), после чего коротко центрифугируют.
Генотипирование микросателлитных локусов Y-хромосомы проводят с помощью капиллярного гель-электрофореза. Продукты ПЦР отдельных локусов объединяют в денатурирующем буфере и разделяют на генетических анализаторах Applied Biosystems (Applied Biosystems Genetic Analyzers). Автоматические ДНК-анализаторы представляют собой автоматизированную систему приборов, предназначенную для определения нуклеотидной последовательности, или размера и количества фрагментов ДНК. Генетический анализатор представляет собой систему капиллярного электрофореза с индуцированной лазером флуоресценцией. Образцы ДНК, меченные с использованием до 4 различных флуоресцентных красителей, собирают и помещают в автодозатор прибора. Электрофорез фрагментов ДНК, меченных красителем, происходит в полимере, в котором они разделяются в соответствии с их размерами. В процессе прохождения через капилляр меченные образцы проходят через специальное окно в капилляре и облучаются. Флуоресцентные красители, связанные с фрагментами ДНК, возбуждаются с помощью лазера и излучают свет при разных длинах волн, характерных для каждого красителя, разделяются в соответствии с длинами волн с помощью спектрографа. Они накапливаются в камере прибора с зарядовой связью (CCD), таким образом, все 4 типа флуоресцентного излучения можно детектировать одновременно. С помощью программного обеспечения световые сигналы накапливаются по интенсивности с использованием фильтров, выбираемых программой (спектральные или волновые фильтры), и хранятся в виде электрических сигналов для последующей обработки данных.
Результаты в итоге представлены в виде разноцветных пиков (каждый из цветов соответствует определенному флуоресцентному красителю) - электрофореграммы, с обозначенным размера продуктов, который устанавливается по стандарту длины, добавляемому при пробоподготовке в каждый образец. Поскольку генотипирование проводится только с использованием образцов ДНК мужчин то наблюдаемые сочетания генотипов разных YSTR отражают их физическое расположение на Y-хромосоме конкретного индивида.
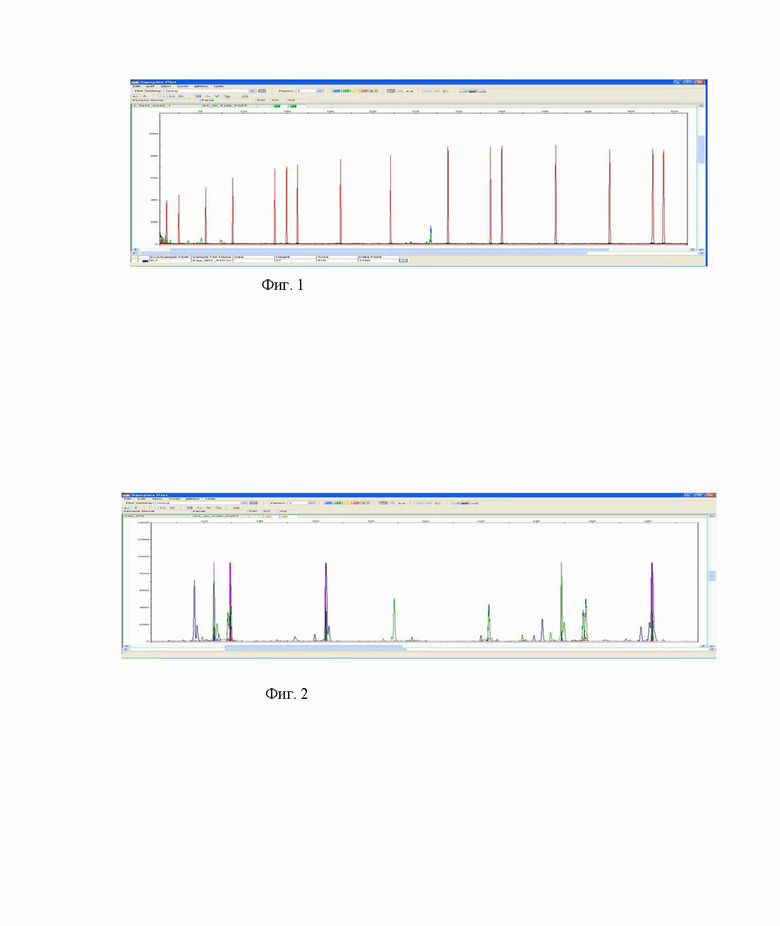
На фигуре 1 приведена электрофореграмма разделения стандарта длины ДНК (GeneScan500-ROX) - красные пики. На фигуре 2 приведена электрофореграмма разделения микросателлитных маркеров Y-хромосомы методом капиллярного гель-электрофореза у одного из индивидов.
Разрабатанный способ обеспечивает возможность получения амплификатов ДНК, содержащих не менее 90% объема специфичных фрагментов ДНК. Неспецифичные фрагменты ДНК допускаются в объеме не превышающем 10% объема (оцениваемого как площадь под пиком соответствующих специфичных фрагментов ДНК). Точность генотипирования не менее 99%. Погрешность измерений длины фрагмента ДНК не более 1 пары нуклеотидов. Воспроизводимость результатов должна обеспечиваться точным соблюдением всех условий геотипирования.
Таким образом, в результате использования предложенного способа генотипирования YSTR-маркеров путем проведения ПЦР и фрагментного анализа исследуемых образцов ДНК, возможно установить комплексный генотип исследуемого образца ДНК человека по 38 микросателлитным маркерам нерекомбинирующей части Y-хромосомы: DYS19, DYS385a, DYS385b, DYS389I, DYS389II, DYS390, DYS391, DYS392, DYS393, DYS437, DYS438, DYS439, DYS442, DYS445, DYS448, DYS449, DYS456, DYS458, DYS460, DYS481, DYS504, DYS505, DYS518, DYS525, DYS533, DYS537, DYS552, DYS570, DYS576, DYS635, DYS 643, YCAIIa, YCAIIb, GATA-H4.1, Y-GATA-A10, GGAAT1B07, DYF387a, DYF387b.
Предлагаемый способ отличается высокой чувствительностью, специфичностью, высокой точностью, обладает высоким дискриминирующим потенциалом и является более экономичным.
Краткое описание чертежей
Фиг. 1 - Электрофореграмма разделения стандарта длины ДНК (GeneScan500-ROX) красные пики.
На фигуре изображена электрофореграмма разделения стандарта длинны для определения размеров продуктов STR-маркеров методом капиллярного электрофореза, проведенного на секвенаторе. Электрофореграмма содержит изображение только стандарт длинны, без добавления исмледуемых образцов ДНК.
Фиг. 2 - Электрофореграмма разделения микросателлитных маркеров Y-хромосомы методом капиллярного гель-электрофореза у одного из индивидов.
На фигуре изображена электрофореграмма разделения продуктов ПЦР исследуемых STR-маркеров входящих в набор, с различными флуоресцентными метками. Электрофореграмма включает продукты ПЦР YSTR-маркеров одного образца принадлежащего мужчине и линейный стандарт длины для определения размеров фрагментов исследуемых маркеров.
Источники информации
1. Jobling М.А. and Tyler-Smith С.New uses for new haplotypes the human Y chromosome, disease and selection // Trends in Genetics. 2000. V. 16. P. 356-362.
2. Jobling M.A., Tyler-Smith C. Fathers and sons: the Y chromosome and human evolution // Trends in Genetics. 1995. V. 11. P. 449 456.
3. Carracedo A., Bar W., Lincoln P., Mayr W., Morling N., Olaisen В., Schneider P., Budowle В., Brinkmann В., Gil P., Holland M., Tully G, Wilson M., DNA Commission of the hiternational Society for Forensic Genetics: guidelines for mitochondrial DNA typing// Forensic Sci. Int. 2000. Vol.110. - P. 79-85.
4. Sanchez, JJ, Phillips, C, Borsting, С et al. (2006) A multiplex assay with 52 single nucleotide polymorphisms for human identification. Electrophoresis/ 2006. V. 27, (9). P. 1713-1724.
5. Butler J.M., Schoske R., Vallone P.M. et al. A novel multiplex for simultaneous amplification of 20 Y chromosome STR markers // Forensic Sci Int. 2002. V.129. P. 10-24.
6. Zhivotovsky L.A., Akhmetova V.L., Fedorova S.A., Zhirkova V.V., Khusnutdininova E.K. Developing STR databases on structured populations: the native South Siberian population versus the Russian population.Forensic Sci Int; Genetics. 2009. V.3. P. 111-116.
7. Степанов B.A., Балановский О.П., Мельников A.B., Лаш-Завада А.Ю., Харьков В.Н., Тяжелова Т.В., Ахметова В.Л., Жукова О.В., Шнейдер Ю.В., Шильникова Н.Н., Боринская С.А., Марусин А.В., Спиридонова М.Г., Симонова К.В., Хитринская И.Ю., Раджабов М.О., Романов А.Г., Штыгашева О.В., Кошель С.М., Балановская Е.В., Рыбакова А.В., Хуснутдинова Э.К., Пузырев В.П., Янковский Н.К. Характеристика популяций Российской Федерации по панели пятнадцати локусов, используемых для ДНК-идентификации и в судебно-медицинской экспертизе // Acta Naturae. 2011. Т. 3. №2. С. 59-71.
8. RU 2558231 «Способ, тест-система и праймеры для определения гаплогрупп Y-хромосомы человека».
9. RU 2528742 «Синтетические олигонуклеотидные праймеры и способ выявления генотипов для идентификации личности с помощью системы микросателлитных ДНК-маркеров Y-хромосомы».
10. RU 2740575 «Набор синтетических олигонуклеотидов для одновременного генотипирования 63 ДНК-маркеров, ассоциированных с группой крови АВО, основными гаплогруппами Y-хромосомы, цветом радужной оболочки глаза, волос, кожи и половой принадлежностью, методом ПЦР с последующей гибридизацией».
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> Научно-исследовательский институт медицинской генетики Федерального
государственного бюджетного научного учреждения Томский национальный
исследовательский медицинский центр Российской академии наук (Research
Institute of Medical Genetics, Tomsk NRMC)
Федеральное государственное бюджетное учреждение науки Институт общей
генетики им. Н.И. Вавилова Российской академии наук (Vavilov Institute of
General Genetics Russian Academy of Sciences)
<120> Набор STR-маркеров Y-хромосомы для определения этно-территориального
происхождения индивида по образцу его ДНК
<130>
<160> 70
<210> 1
<211> 19
<212> DNA
<213> Homo sapiens
<400> 1
agcatgggtg acagagcta
<210> 2
<211> 20
<212> DNA
<213> Homo sapiens
<400> 2
tgggatgcta ggtaaagctg
<210> 3
<211> 25
<212> DNA
<213> Homo sapiens
<400> 3 26
ccaactctca tctgtattat ctatg
<210> 4
<211> 20
<212> DNA
<213> Homo sapiens
<400> 4
tcttatctcc acccaccaga
<210> 5
<211> 23
<212> DNA
<213> Homo sapiens
<400> 5
tatattttac acatttttgg gcc
<210> 6
<211> 23
<212> DNA
<213> Homo sapiens
<400> 6
tgacagtaaa atgaacacat tgc
<210> 7
<211> 23
<212> DNA
<213> Homo sapiens
<400> 7
ctattcattc aatcatacac cca
<210> 8
<211> 20 27
<212> DNA
<213> Homo sapiens
<400> 8
gattctttgt ggtgggtctg
<210> 9
<211> 25
<212> DNA
<213> Homo sapiens
<400> 9
tcattaatct agcttttaaa aacaa
<210> 10
<211> 20
<212> DNA
<213> Homo sapiens
<400> 10
agacccagtt gatgcaatgt
<210> 11
<211> 24
<212> DNA
<213> Homo sapiens
<400> 11
gtggtcttct acttgtgtca atac
<210> 12
<211> 22
<212> DNA
<213> Homo sapiens
<400> 12 28
aactcaagtc caaaaaatga gg
<210> 13
<211> 21
<212> DNA
<213> Homo sapiens
<400> 13
ctactgagtt tctgttatag t
<210> 14
<211> 19
<212> DNA
<213> Homo sapiens
<400> 14
atggcatgta gtgaggaca
<210> 15
<211> 20
<212> DNA
<213> Homo sapiens
<400> 15
gactatgggc gtgagtgcat
<210> 16
<211> 23
<212> DNA
<213> Homo sapiens
<400> 16
gagaccctgt cattcacaga tga
<210> 17
<211> 23 29
<212> DNA
<213> Homo sapiens
<400> 17
ccaaaattag tggggaatag ttg
<210> 18
<211> 21
<212> DNA
<213> Homo sapiens
<400> 18
gatcacccag ggtctggagt t
<210> 19
<211> 24
<212> DNA
<213> Homo sapiens
<400> 19
tcgagttgtt atggttttag gtct
<210> 20
<211> 22
<212> DNA
<213> Homo sapiens
<400> 20
gtggcttgga attcttttac cc
<210> 21
<211> 21
<212> DNA
<213> Homo sapiens
<400> 21 30
ccccaagtcc ccaaagtgtg t
<210> 22
<211> 23
<212> DNA
<213> Homo sapiens
<400> 22
cactcattga ttgatgggcg ttt
<210> 23
<211> 22
<212> DNA
<213> Homo sapiens
<400> 23
agttaagagc cccaccttcc tg
<210> 24
<211> 23
<212> DNA
<213> Homo sapiens
<400> 24
ttttggtggc ataatctcag ctc
<210> 25
<211> 20
<212> DNA
<213> Homo sapiens
<400> 25
tgggagaggc aaggatccaa
<210> 26
<211> 22 31
<212> DNA
<213> Homo sapiens
<400> 26
ccagaccggc cagaaatatg ac
<210> 27
<211> 25
<212> DNA
<213> Homo sapiens
<400> 27
ttttccctta acttgtgtga ttttt
<210> 28
<211> 25
<212> DNA
<213> Homo sapiens
<400> 28
aggttggaca acaagagtaa gacag
<210> 29
<211> 18
<212> DNA
<213> Homo sapiens
<400> 29
ggaccttgtg ataatgta
<210> 30
<211> 21
<212> DNA
<213> Homo sapiens
<400> 30 32
gttttgggct gagttgatgg g
<210> 31
<211> 23
<212> DNA
<213> Homo sapiens
<400> 31
agcaacagga atgaaactcc aat
<210> 32
<211> 18
<212> DNA
<213> Homo sapiens
<400> 32
ggagggtggg cgtggtgg
<210> 33
<211> 22
<212> DNA
<213> Homo sapiens
<400> 33
gaggaatctg acacctctga ca
<210> 34
<211> 25
<212> DNA
<213> Homo sapiens
<400> 34
gtccatatca tctatcctct gccta
<210> 35
<211> 20 33
<212> DNA
<213> Homo sapiens
<400> 35
aggaatgtgg ctaacgctgt
<210> 36
<211> 20
<212> DNA
<213> Homo sapiens
<400> 36
acagctcacc agaaggttgc
<210> 37
<211> 20
<212> DNA
<213> Homo sapiens
<400> 37
tctacaccac tgtgccaagc
<210> 38
<211> 19
<212> DNA
<213> Homo sapiens
<400> 38
ggcaacagag caaccctct
<210> 39
<211> 20
<212> DNA
<213> Homo sapiens
<400> 39 34
tctggcgaag taacccaaac
<210> 40
<211> 21
<212> DNA
<213> Homo sapiens
<400> 40
tcgagtcagt tcaccagaag g
<210> 41
<211> 20
<212> DNA
<213> Homo sapiens
<400> 41
ggcaacacaa gtgaaactgc
<210> 42
<211> 21
<212> DNA
<213> Homo sapiens
<400> 42
tcagctctta ccatgggtga t
<210> 43
<211> 20
<212> DNA
<213> Homo sapiens
<400> 43
attcacacca ttgcactcca
<210> 44
<211> 20 35
<212> DNA
<213> Homo sapiens
<400> 44
gaggcggagc ttttagtgag
<210> 45
<211> 20
<212> DNA
<213> Homo sapiens
<400> 45
gacccactgg cattcaaatc
<210> 46
<211> 21
<212> DNA
<213> Homo sapiens
<400> 46
tgctcccttt ctttgtagac g
<210> 47
<211> 24
<212> DNA
<213> Homo sapiens
<400> 47
catctaacat ctttgtcatc tacc
<210> 48
<211> 23
<212> DNA
<213> Homo sapiens
<400> 48 36
tgatcagttc ttaactcaac caa
<210> 49
<211> 20
<212> DNA
<213> Homo sapiens
<400> 49
ggtctccaat tccatccaga
<210> 50
<211> 20
<212> DNA
<213> Homo sapiens
<400> 50
tggaacatgc ccattaatca
<210> 51
<211> 20
<212> DNA
<213> Homo sapiens
<400> 51
ccatagtgcc gaggtcaagt
<210> 52
<211> 19
<212> DNA
<213> Homo sapiens
<400> 52
aacacctgat gcctggttg
<210> 53
<211> 22 37
<212> DNA
<213> Homo sapiens
<400> 53
gaactgtcta caatggctca cg
<210> 54
<211> 26
<212> DNA
<213> Homo sapiens
<400> 54
tcagcatagt caagaaacca gacaac
<210> 55
<211> 19
<212> DNA
<213> Homo sapiens
<400> 55
tgggctgagg agttcaatc
<210> 56
<211> 23
<212> DNA
<213> Homo sapiens
<400> 56
ggcagtctca tttcctggag atg
<210> 57
<211> 26
<212> DNA
<213> Homo sapiens
<400> 57 38
agtgtctcac ttcaagcacc aagcac
<210> 58
<211> 28
<212> DNA
<213> Homo sapiens
<400> 58
acatttttcc aactgtgaat tttgctgc
<210> 59
<211> 20
<212> DNA
<213> Homo sapiens
<400> 59
aagccatgcc tggttaaact
<210> 60
<211> 21
<212> DNA
<213> Homo sapiens
<400> 60
tgtaaccaaa caccacccat t
<210> 61
<211> 23
<212> DNA
<213> Homo sapiens
<400> 61
tgtcaaaatt taacccacaa tca
<210> 62
<211> 24 39
<212> DNA
<213> Homo sapiens
<400> 62
gcagtctttc accataaggt tagc
<210> 63
<211> 20
<212> DNA
<213> Homo sapiens
<400> 63
atgctgagga gaatttccaa
<210> 64
<211> 27
<212> DNA
<213> Homo sapiens
<400> 64
gctattcatc catctaatct atccatt
<210> 65
<211> 28
<212> DNA
<213> Homo sapiens
<400> 65
cctgccatct ctatttatct tgcatata
<210> 66
<211> 24
<212> DNA
<213> Homo sapiens
<400> 66 40
ataaatggag atagtgggtg gatt
<210> 67
<211> 23
<212> DNA
<213> Homo sapiens
<400> 67
cattaaccat agcatttctt tcc
<210> 68
<211> 22
<212> DNA
<213> Homo sapiens
<400> 68
gaagtagagt ggaatggact gc
<210> 69
<211> 20
<212> DNA
<213> Homo sapiens
<400> 69
gcctgggtga cagagctaga
<210> 70
<211> 21
<212> DNA
<213> Homo sapiens
<400> 70
gccacagtgt gagaagtgtg a 41
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Тест-система и способ выявления делеций длинного плеча 6 хромосомы | 2021 |
|
RU2770892C1 |
| Тест-система и способ выявления делеций гена SESN1 | 2021 |
|
RU2772504C1 |
| СИНТЕТИЧЕСКИЕ ОЛИГОНУКЛЕОТИДНЫЕ ПРАЙМЕРЫ И СПОСОБ ВЫЯВЛЕНИЯ ГЕНОТИПОВ ДЛЯ ИДЕНТИФИКАЦИИ ЛИЧНОСТИ С ПОМОЩЬЮ СИСТЕМЫ МИКРОСАТЕЛЛИТНЫХ ДНК-МАРКЕРОВ Y-ХРОМОСОМЫ | 2012 |
|
RU2528742C2 |
| СПОСОБЫ И КОМПОЗИЦИИ ДЛЯ ДНК-ПРОФИЛИРОВАНИЯ | 2015 |
|
RU2752700C2 |
| Набор для выявления коронавируса SARS-CoV-2 | 2021 |
|
RU2765497C1 |
| СПОСОБ ГЕНЕТИЧЕСКОГО INDEL-ТИПИРОВАНИЯ ШТАММОВ BRUCELLA MELITENSIS | 2019 |
|
RU2732425C1 |
| БИОЛОГИЧЕСКИЙ МИКРОЧИП С НАБОРОМ ПРАЙМЕРОВ ДЛЯ АНАЛИЗА ПОЛИМОРФИЗМА В ГЕНАХ AB0, HLA-DQA1, AMEL, DARC, NAT2 | 2014 |
|
RU2582216C2 |
| Мультиплексный набор маркеров для проведения ассоциативного анализа регуляторных полиморфных маркеров генов подверженности преэклампсии | 2020 |
|
RU2757321C1 |
| НАБОР ОЛИГОНУКЛЕОТИДНЫХ ПРАЙМЕРОВ И ЗОНДОВ ДЛЯ ГЕНОТИПИРОВАНИЯ ПОЛИМОРФНЫХ ЛОКУСОВ ДНК, АССОЦИИРОВАННЫХ С РИСКОМ РАЗВИТИЯ СПОРАДИЧЕСКОЙ ФОРМЫ БОЛЕЗНИ АЛЬЦГЕЙМЕРА В РОССИЙСКИХ ПОПУЛЯЦИЯХ | 2014 |
|
RU2600874C2 |
| Набор для определения копийности гена АРР в геноме человека | 2021 |
|
RU2789799C1 |
Изобретение относится к биотехнологии, молекулярной генетике и может быть использовано для выявления генотипов для идентификации личности и определения этнической принадлежности человека по образцу его ДНК с помощью системы микросателлитных ДНК-маркеров Y-хромосомы. Представлен набор генетических маркеров. Набор содержит синтетические олигонуклеотидные праймеры, представляющие собой нуклеотидные последовательности SEQ ID NO: 1-70 и определяющие 38 коротких тандемных повторов, локализованных на нерекомбинирующем участке Y-хромосомы (DYS19, DYS385a, DYS385b, DYS389I, DYS389II, DYS390, DYS391, DYS392, DYS393, DYS437, DYS438, DYS439, DYS442, DYS445, DYS448, DYS449, DYS456, DYS458, DYS460, DYS481, DYS504, DYS505, DYS518, DYS525, DYS533, DYS537, DYS552, DYS570, DYS576, DYS635, DYS 643, YCAIIa, YCAIIb, GATA-H4.1, Y-GATA-A10, GGAAT1B07, DYF387a, DYF387b). Предложен способ выявления генотипов для идентификации личности в российской популяции с использованием вышеуказанных праймеров. Выбранные микросателлитные маркеры являются достаточно высокоинформативными для большинства популяционных выборок. 2 н.п. ф-лы, 2 ил.
1. Набор синтетических олигонуклеотидных праймеров, предназначенный для ДНК-идентификации индивида, состоящий из пар синтетических олигонуклеотидных праймеров с нуклеотидными последовательностями SNP ID NO: 1-20, отличающийся тем, что он дополнительно содержит пары синтетических олигонуклеотидных праймеров с нуклеотидными последовательностями SNP ID NO: 21-70, при этом синтетические олигонуклеотидные праймеры имеют следующий нуклеотидный состав:
DYS385 (SNP ID NO: 1) F: 5'-TET-AGCATGGGTGACAGAGCTA-3'
DYS385 (SNP ID NO: 2) R: 5'-TGGGATGCTAGGTAAAGCTG
DYS389 (SNP ID NO: 3) F: 5'-HEX-CCAACTCTCATCTGTATTATCTATG
DYS389 (SNP ID NO: 4) R: 5'-TCTTATCTCCACCCACCAGA
DYS390 (SNP ID NO: 5) F: 5'-TET-TATATTTTACACATTTTTGGGCC
DYS390 (SNP ID NO: 6) R: 5'-TGACAGTAAAATGAACACATTGC
DYS391 (SNP ID NO: 7) F: 5'-TET-СТАТТСАТТСААТСАТАСАСССА
DYS391 (SNP ID NO: 8) R: GATTCTTTGTGGTGGGTCTG
DYS392 (SNP ID NO: 9) F: 5'-FAM-TCATTAATCTAGCTTTTAAAAACAA
DYS392 (SNP ID NO: 10) R: 5'-AGACCCAGTTGATGCAATGT
DYS393 (SNP ID NO: 11) F: 5'-FAM-GTGGTCTTCTACTTGTGTCAATAC
DYS393 (SNP ID NO: 12) R: 5'-AACTCAAGTCCAAAAAATGAGG
DYS394 (SNP ID NO: 13) F: 5'-HEX-CTACTGAGTTTCTGTTATAGT
DYS394 (SNP ID NO: 14) R: 5'-ATGGCATGTAGTGAGGACA
DYS437 (SNP ID NO: 15) F: 5'-FAM-GACTATGGGCGTGAGTGCAT-3'
DYS437 (SNP ID NO: 16) R: 5'-GAGACCCTGTCATTCACAGATGA
DYS438 (SNP ID NO: 17) F: 5'-FAM-CCAAAATTAGTGGGGAATAGTTG-3'
DYS438 (SNP ID NO: 18) R: 5'-GATCACCCAGGGTCTGGAGTT-3'
DYS439 (SNP ID NO: 19) F: 5'-TET-TCGAGTTGTTATGGTTTTAGGTCT-3'
DYS439 (SNP ID NO: 20) R: 5'-GTGGCTTGGAATTCTTTTACCC-3'
DYS442 (SNP ID NO: 21) F: 5'- HEX-CCCCAAGTCCCCAAAGTGTGT-3'
DYS442 (SNP ID NO: 22) R: 5'-CACTCATTGA TTGATGGGCGTTT-3'
DYS445 (SNP ID NO: 23) F: 5'- FAM-AGTTAAGAGCCCCACCTTCCTG-3'
DYS445 (SNP ID NO: 24) R: 5'-TTTTGGTGGCATAATCTCAGCTC-3'
DYS448 (SNP ID NO: 25) F: 5'- FAM-TGGGAGAGGCAAGGATCCAA-3'
DYS448 (SNP ID NO; 26) R: 5'-CCAGACCGGCCAGAAATATGAC-3'
DYS449 (SNP ID NO: 27) F: 5'- FAM-TTTTCCCTTAACTTGTGTGATTTTT-3'
DYS449 (SNP ID NO: 28) R: 5'-AGGTTGGACAACAAGAGTAAGACAG-3'
DYS456 (SNP ID NO: 29) F: 5'- FAM-GGACCTTGTGATAATGTA-3'
DYS456 (SNP ID NO: 30) R: 5'-GTTTTGGGCTGAGTTGATGGG-3'
DYS458 (SNP ID NO: 31) F: 5'- FAM-AGCAACAGGAATGAAACTCCAAT-3'
DYS458 (SNP ID NO: 32) R: 5'-GGAGGGTGGGCGTGGTGG-3'
DYS460 (SNP ID NO: 33) F: 5'- ROX-GAGGAATCTGACACCTCTGACA-3'
DYS460 (SNP ID NO: 34) R: 5'-GTCCATATCATCTATCCTCTGCCTA-3'
DYS481 (SNP ID NO: 35) F: 5'- JOE-AGGAATGTGGCTAACGCTGT-3'
DYS481 (SNP ID NO: 36) R: 5'-ACAGCTCACCAGAAGGTTGC-3'
DYS504 (SNP ID NO: 37) F: 5'- HEX-TCTACACCACTGTGCCAAGC-3'
DYS504 (SNP ID NO: 38) R: 5'-GGCAACAGAGCAACCCTCT-3'
DYS505 (SNP ID NO: 39) F: 5'- FAM-TCTGGCGAAGTAACCCAAAC-3'
DYS505 (SNP ID NO: 40) R: 5'-TCGAGTCAGTTCACCAGAAGG-3'
DYS518 (SNP ID NO: 41) F: 5'- JOE-GGCAACACAAGTGAAACTGC-3'
DYS518 (SNP ID NO: 42) R: 5'-TCAGCTCTTACCATGGGTGAT-3'
DYS525 (SNP ID NO: 43) F: 5'- HEX-ATTCACACCATTGCACTCCA-3'
DYS525 (SNP ID NO: 44) R: 5'-GAGGCGGAGCTTTTAGTGAG-3'
DYS531 (SNP ID NO: 45) F: 5'- FAM-GACCCACTGGCATTCAAATC-3'
DYS531 (SNP ID NO: 46) R: 5'-TGCTCCCTTTCTTTGTAGACG-3'
DYS533 (SNP ID NO: 47) F: 5'- JOE-CATCTAACATCTTTGTCATCTACC-3'
DYS533 (SNP ID NO: 48) R: 5'-TGATCAGTTCTTAACTCAACCAA-3'
DYS537 (SNP ID NO: 49) F: 5'- HEX-GGTCTCCAATTCCATCCAGA-3'
DYS537 (SNP ID NO: 50) R: 5'-TGGAACATGCCCATTAATCA-3'
DYS552 (SNP ID NO: 51) F: 5'- FAM-CCATAGTGCCGAGGTCAAGT-3'
DYS552 (SNP ID NO: 52) R: 5'-AACACCTGATGCCTGGTTG-3'
DYS570 (SNP ID NO: 53) F: 5'- ROX-GAACTGTCTACAATGGCTCACG-3'
DYS570 (SNP ID NO: 54) R: 5' -TCAGCATAGTCAAGAAACCAGACAAC-3'
DYS576 (SNP ID NO: 55) F: 5'- ROX-TGGGCTGAGGAGTTCAATC-3'
DYS576 (SNP ID NO: 56) R: 5'-GGCAGTCTCATTTCCTGGAGATG
DYS635 (SNP ID NO: 57) F: 5'- FAM-AGTGTCTCACTTCAAGCACCAAGCAC-3'
DYS635 (SNP ID NO: 58) R: 5'-ACATTTTTCCAACTGTGAATTTTGCTGC-3'
DYS643 (SNP ID NO: 59) F: 5'- FAM-AAGCCATGCCTGGTTAAACT-3'
DYS643 (SNP ID NO: 60) R: 5'-TGTAACCAAACACCACCCATT-3'
YCAII (SNP ID NO: 61) F: 5'- HEX-TGTCAAAATTTAACCCACAATCA-3'
YCAII (SNP ID NO: 62) R: 5'-GCAGTCTTTCACCATAAGGTTAGC-3'
GATA H4.1 (SNP ID NO: 63) F: 5'- HEX-ATGCTGAGGAGAATTTCCAA-3'
GATA H4.1 (SNP ID NO: 64) R: 5'-GCTATTCATCCATCTAATCTATCCATT-3'
Y-GATA-A10 (SNP ID NO: 65) F: 5'- NED-CCTGCCATCTCTATTTATCTTGCATATA-3'
Y-GATA-A10 (SNP ID NO: 66) R: 5'-ATAAATGGAGATAGTGGGTGGATT-3'
GGAAT1B07 (SNP ID NO: 67) F: 5'- NED-CATTAACCATAGCATTTCTTTCC-3'
GGAAT1B07 (SNP ID NO: 68) R: 5'-GAAGTAGAGTGGAATGGACTGC-3'
DYF387S1 (SNP ID NO: 69) F: 5'- FAM-GCCTGGGTGACAGAGCTAGA-3'
DYF387S1 (SNP ID NO: 70) R: 5'-GCCACAGTGTGAGAAGTGTGA-3'.
2. Способ выявления генотипов для идентификации личности с помощью системы микросателлитных ДНК-маркеров Y-хромосомы, отличающийся тем, что используют набор праймеров по п. 1, при этом проводят генотипирование 38 коротких тандемных повторов, локализованных на нерекомбинирующем участке Y-хромосомы (YSTR), на генетическом ДНК-анализаторе с помощью капиллярного гель-электрофореза, с применением метода ПЦР и фрагментного анализа флуоресцентно меченых ДНК-ампликонов, используют образцы ДНК мужчин, выделенную из любого биологического материала в соответствии со стандартными методами, обеспечивающими качественную очистку ДНК от белков и примесей и предотвращающими деградацию материала в ходе выделения, при проведении капиллярного электрофореза фрагменты, синтезированные в ходе ПЦР, имеющие флуоресцентную метку ТЕХ (4,7,2',7'-тетрахлоро-6-карбоксифлуоресцеин) в диапазоне длин 360-420 п.о., соответствуют аллелям 9-23 тандемных повтора в локусе DYS385, фрагменты, имеющие флуоресцентную метку ТЕТ в диапазоне длин 196-224 п.о., соответствуют аллелям 19-26 тандемных повторов в локусе DYS390, фрагменты, имеющие флуоресцентную метку ТЕТ в диапазоне длин 277-289 п.о., соответствуют аллелям 9-12 тандемных повторов в локусе DYS391, фрагменты, имеющие флуоресцентную метку ТЕТ в диапазоне длин 232-260 п.о., соответствуют аллелям 8-15 тандемных повторов в локусе DYS439, фрагменты, имеющие флуоресцентную метку FAM (6-карбоксифлуоресцсин) в диапазоне длин 243-259 п.о., соответствуют аллелям 10-15 тандемных повторов в локусе DYS392, фрагменты, имеющие флуоресцентную метку FAM в диапазоне длин 116-128 п.о., соответствуют аллелям 12-15 тандемных повторов в локусе DYS393, фрагменты, имеющие флуоресцентную метку FAM в диапазоне длин 174-190 п.о., соответствуют аллелям 6-10 тандемных повторов в локусе DYS437, фрагменты, имеющие флуоресцентную метку FAM в диапазоне длин 311-326 п.о., соответствуют аллелям 9-12 тандемных повторов в локусе DYS438, фрагменты, имеющие флуоресцентную метку FAM в диапазоне длин 251-263 п.о., соответствуют аллелям 9-12 тандемных повторов в локусе DYS445, фрагменты, имеющие флуоресцентную метку FAM в диапазоне длин 307-343 п.о., соответствуют аллелям 18-24 тандемных повторов в локусе DYS448, фрагменты, имеющие флуоресцентную метку FAM в диапазоне длин 264-313 п.о., соответствуют аллелям 25-37 тандемных повторов в локусе DYS449, фрагменты, имеющие флуоресцентную метку FAM в диапазоне длин 141-157 п.о., соответствуют аллелям 17-17 тандемных повторов в локусе DYS456, фрагменты, имеющие флуоресцентную метку FAM в диапазоне длин 106-156 п.о., соответствуют аллелям 12-24 тандемных повторов в локусе DYS458, фрагменты, имеющие флуоресцентную метку FAM в диапазоне длин 165-181 п.о., соответствуют аллелям 11-15 тандемных повторов в локусе DYS505, фрагменты, имеющие флуоресцентную метку FAM в диапазоне длин 102-127 п.о., соответствуют аллелям 8-14 тандемных повторов в локусе DYS531, фрагменты, имеющие флуоресцентную метку FAM в диапазоне длин 236-256 п.о., соответствуют аллелям 7-12 тандемных повторов в локусе DYS552. фрагменты, имеющие флуоресцентную метку FAM в диапазоне длин 238-274 п.о., соответствуют аллелям 17-26 тандемных повторов в локусе DYS635, фрагменты, имеющие флуоресцентную метку FAM в диапазоне длин 123-158 п.о., соответствуют аллелям 7-14 тандемных повторов в локусе DYS643. фрагменты, имеющие флуоресцентную метку FAM в диапазоне длин 240-272 п.о., соответствуют аллелям 27-35 тандемных повторов в локусе DYF387, фрагменты, имеющие флуоресцентную метку HEX (4,7,2',4',5',7'-гексахлоро-6-карбоксифлуоресцеин) в диапазоне длин 190-206 п.о., соответствуют аллелям 13-17 тандемных повторов в локусе DYS394, фрагменты, имеющие флуоресцентную метку HEX в диапазоне длин 242-262 п.о., соответствуют аллелям 7-12 тандемных повторов в локусе DYS389I. фрагменты, имеющие флуоресцентную метку HEX в диапазоне длин 360-382 п.о., соответствуют локусу DYS389II, аллели локуса DYS389II определяют вычитанием из значения длины фрагмента диапазона 360-382 значения длины фрагмента DYS389I, получаемый диапазон 110-134 п.о. соответствует 14-20 тандемным повторам, фрагменты, имеющие флуоресцентную метку HEX в диапазоне длин 298-322 п.о., соответствуют аллелям 10-16 тандемных повторов в локусе DYS442, фрагменты, имеющие флуоресцентную метку HEX в диапазоне длин 265-296 п.о., соответствуют аллелям 12-20 тандемных повторов в локусе DYS504, фрагменты, имеющие флуоресцентную метку HEX в диапазоне длин 305-321 п.о., соответствуют аллелям 9-13 тандемных повторов в локусе DYS525, фрагменты, имеющие флуоресцентную метку HEX в диапазоне длин 155-179 п.о., соответствуют аллелям 7-13 тандемных повторов в локусе DYS537, фрагменты, имеющие флуоресцентную метку HEX в диапазоне длин 147-165 п.о., соответствуют аллелям 17-26 тандемных повторов в локусе YCAII, фрагменты, имеющие флуоресцентную метку HEX в диапазоне длин 134-152 п.о., соответствуют аллелям 9-14 тандемных повторов в локусе Y-GATA-H4.1, фрагменты, имеющие флуоресцентную метку ROX (карбокси-Х-родамин) в диапазоне длин 102-122 п.о., соответствуют аллелям 8-13 тандемных повторов в локусе DYS460, фрагменты, имеющие флуоресцентную метку ROX в диапазоне длин 242-282 п.о., соответствуют аллелям 13-23 тандемных повторов в локусе DYS570, фрагменты, имеющие флуоресцентную метку ROX в диапазоне длин 168-204 п.о., соответствуют аллелям 13-22 тандемных повторов в локусе DYS576, фрагменты, имеющие флуоресцентную метку JOE в диапазоне длин 117-157 п.о., соответствуют аллелям 189-32 тандемных повторов в локусе DYS481, фрагменты, имеющие флуоресцентную метку JOE (6-карбокси-4',5'-дихлор-2',7'-диметоксифлуоресцеин) в диапазоне длин 252-296 п.о., соответствуют аллелям 25-36 тандемных повторов в локусе DYS518, фрагменты, имеющие флуоресцентную метку JOE в диапазоне длин 200-224 п.о., соответствуют аллелям 9-15 тандемных повторов в локусе DYS533; фрагменты, имеющие флуоресцентную метку NED, соответствуют локусу Y-GATA-A10 или GGAAT1B07.
| СИНТЕТИЧЕСКИЕ ОЛИГОНУКЛЕОТИДНЫЕ ПРАЙМЕРЫ И СПОСОБ ВЫЯВЛЕНИЯ ГЕНОТИПОВ ДЛЯ ИДЕНТИФИКАЦИИ ЛИЧНОСТИ С ПОМОЩЬЮ СИСТЕМЫ МИКРОСАТЕЛЛИТНЫХ ДНК-МАРКЕРОВ Y-ХРОМОСОМЫ | 2012 |
|
RU2528742C2 |
| СТЕПАНОВ В.А | |||
| и др., Характеристика популяций Российской Федерации по панели пятнадцати локусов, используемых для ДНК-идентификации и в судебномедицинской экспертизе, том 3 N, 2 (9) 2011, Acta naturae, с | |||
| Устройство для охлаждения водою паров жидкостей, кипящих выше воды, в применении к разделению смесей жидкостей при перегонке с дефлегматором | 1915 |
|
SU59A1 |
Авторы
Даты
2023-09-29—Публикация
2021-11-15—Подача