Изобретение относится к области молекулярной биологии, биотехнологии, судебной медицины и криминалистики, генетическим исследованиям в области идентификации человека и используется для определения полной последовательности митохондриальной ДНК человека. Изобретение представляет собой набор оригинальных синтетических олигонуклеотидиых прайм еров для проведения мультиплексной полимеразной цепной реакции (ГЩР). Изобретение дает возможность получения перекрывающихся фрагментов митохондриальной ДНК из образцов геномной ДНК человека, в том числе в случае, когда ДНК в исследуемом образце находится в деградированном виде.
Уровень техники
Митохондриальная ДНК является крайне удобным маркером и успешно используется в области судебной медицины и криминалистике [1, 2]. При работе с деградированной и древней ДНК, митохондриальная ДНК (мтДНК) имеет явные преимущества перед ядерной за счет ряда факторов, основным из которых является ее многокопийность (до нескольких тысяч, в отличие от двух копий ядерных генов). Это значительно повышает вероятность сохранности фрагментов ДНК и успешной амплификации в ходе ПЦР, что чрезвычайно удобно в ситуациях, когда количество ДНК в исследуемом образце очень ограничено, и дает возможность генотипирования мтДНК даже в образцах с высокой степенью деградации ДНК. Кроме того, из многих типов образцов, используемых в криминалистических исследованиях, мтДНК выделяется гораздо лучше, чем ядерная, что также является ключевым преимуществом этой ДНК для исследований образцов с мест преступлений. К подобным типам образцов относятся волосы, для которых показано значительное количество мтДНК по сравнению с ядерной (после кератинизации волосяного стержня, ядра его клеток разрушаются, тогда как митохондрии сохраняются), причем на уровень различий также влияет область волоса, взятая для исследований [3]. Отпечатки пальцев, полученные с использованием как магнитных, так и немагнитных черных дактилопорошков, также демонстрируют большее количество мтДНК по сравнению с ядерной [3|. Таким образом, данный тип генетического материала является крайне актуальным для криминалистических исследований на образцах, продолжительное время пребывавших в условиях, способствующих деградации ДНК. Явление наследования мтДНК единым локусом по материнской линии, кроме одного случая отцовского вклада у мужчины с митохондриальной миопатией [4], также используется в криминалистических экспертизах, к примеру, в случаях, где сравнение неизвестных образцов с ближайшими родственниками по каким-либо причинам невозможно, проводится сравнение с родственниками по материнской линии. МтДНК также широко применяется при идентификации лиц, пострадавших от катастроф и природных бедствий, а также для исследовании улик преступлений, произошедших многие годы назад, так как мтДНК можно выделить в случаях, когда тело пострадавшего оказалось сильно повреждено или возраст образцов тканей пострадавшего исчисляется годами (к примеру, улики из старых, нераскрытых преступлений).
В настоящее время разработаны методики успешного использования анализа полиморфизма митохондриальной ДНК для судебно-медицинских целей, основанные на определении отдельных вариантов митохондриальной ДНК путем анализа длин реетрикционных фрагментов, а также путем секвенирования по методу Сенгера. Однако, также, существует ряд методов, позволяющих успешно реконструировать полные последовательности мтДНК изучаемых образцов с высокой степенью деградации. Метод мультиплексной ПЦР широко применяется как в популяционных и криминалистических исследованиях, так и при изучении деградированной ДНК древних, музейных и исторических образцов не человеческого происхождения [5]. Ранее была представлена методика, в которой разработано два пуша праймеров с взаимопересекающимися ампликонами размером 144-230 п.о. для изготовления библиотек секвенирования и последующей реконструкции полных последовательностей человеческой мтДНК [6]. Методы гибридизации на ДНК- или РНК зондах, изготовленных из недеградированной ДНК, также являются актуальными, причем зонды могут быть как видоспецифичными и изготовленными из ДНК хорошей сохранности того же вида, что и изучаемый образец, так и кладоспецифичными, с использованием ДНК близкородственных видов для изготовления зондов [7, 8]. Для человеческой ДНК по этому принципу были изготовлены ДНК-зонды для гибридизации длиной 150-850 и.о. на мт последовательность [9]. Подобные методы успешно применяются в первую очередь для отделения деградированной целевой ДНК от контаминирующей ДНК в той же пробе. Технологии высокопроизводительного параллельного секвенирования нового поколения также применяются в сочетании с данной методикой путем насыщения пулированной библиотеки секвенирования целевой ДНК с последующим получением данных для большого количества индивидуальных последовательностей [10]. Методика обогащения целевой ДНК также была разработана для полной мтДНК Неандертальца [11] и впоследствии модифицирована для контрольного участка мтДНК человека с размером ампликонов примерно 70 п.н. [12]. Полная либо частичная последовательность мтДНК может быть также получена в ходе применения секвенирования нового поколения геномных библиотек изучаемых образцов. На сегодняшний день наборов, нацеленных на работу именно с митохондриальной ДНК не очень много (Табл. 1). Наборы Mitochondrial DNA Ilypervariable Region I and II PGR Amplification Kit (Norgen Biotek Corp) и LINEAR ARRAY mtDNA HVI/HVII Region-Sequence Typing Kit (Merck, бывшая Sigma-Aldrich) ориентированы на амплификацию наиболее вариабельных регионов митохондриального генома, гипервариабельных участков Д-петли, для человека. Также на человекую мтДНК ориентирован и набор REPLI-g Mitochondrial DNA Kit (Qiagen), работающий на насыщение образца геномной ДНК мтДНК путем амплификации собственным набором праймеров с использованием крайне продуктивной полимеразы REPLI-g DNA Polymerase. Имеющиеся в настоящее время наборы олигонуклеотидов, используемые в российских и зарубежных разработках, предназначены для анализа достаточно больших фрагментов мтДНК (более 200-300 п.н.). ДНК такого размера может быть недоступна в случае анализа образцов биологического материала низкого качества. Кроме того, уже разработанные олигонуклеотиды в большинстве своем не учитывают популяционную специфику полиморфизма мтДНК населения России Федерации (большое число этнических групп, специфические для различных этнических групп полиморфизмы, наличии популяций как европеоидного, так и монголоидного и смешанного происхождения). Для устранения указанных недостатков возникает необходимость в разработке информативного набора реагентов для анализа высокополиморфных популяций Евразии, а также адаптация такого набора для специфики используемых в криминалистических экспертизах образцов биологического материала, где ДНК сильно деградирована или содержится в малом количестве.
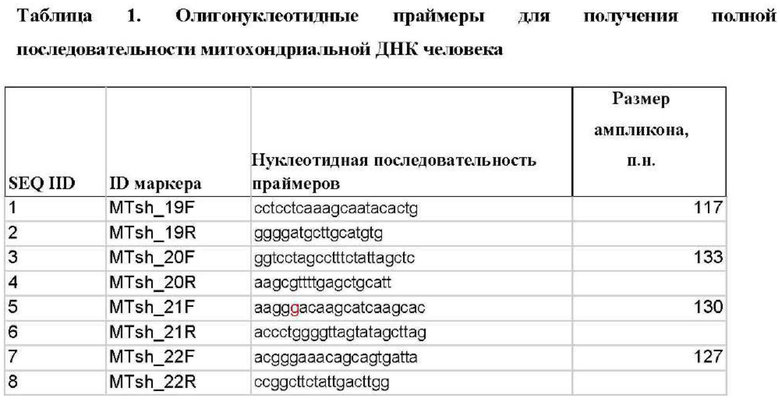
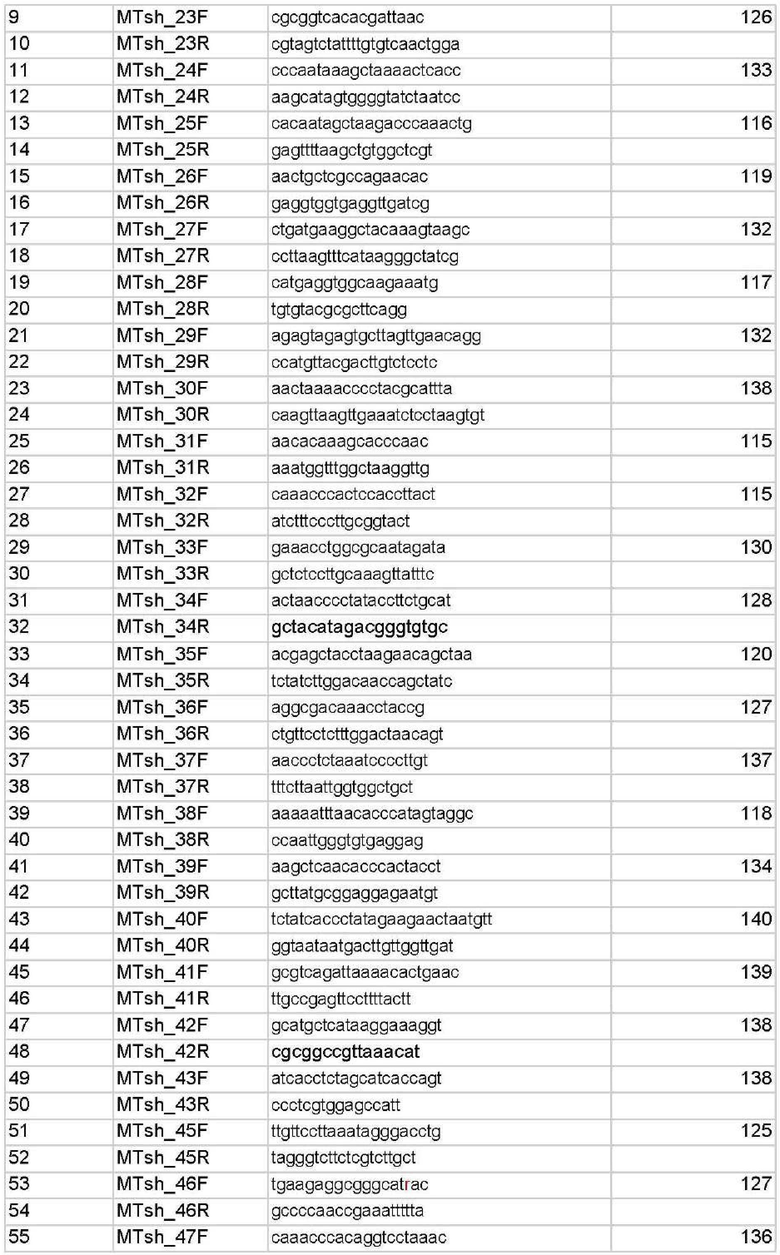
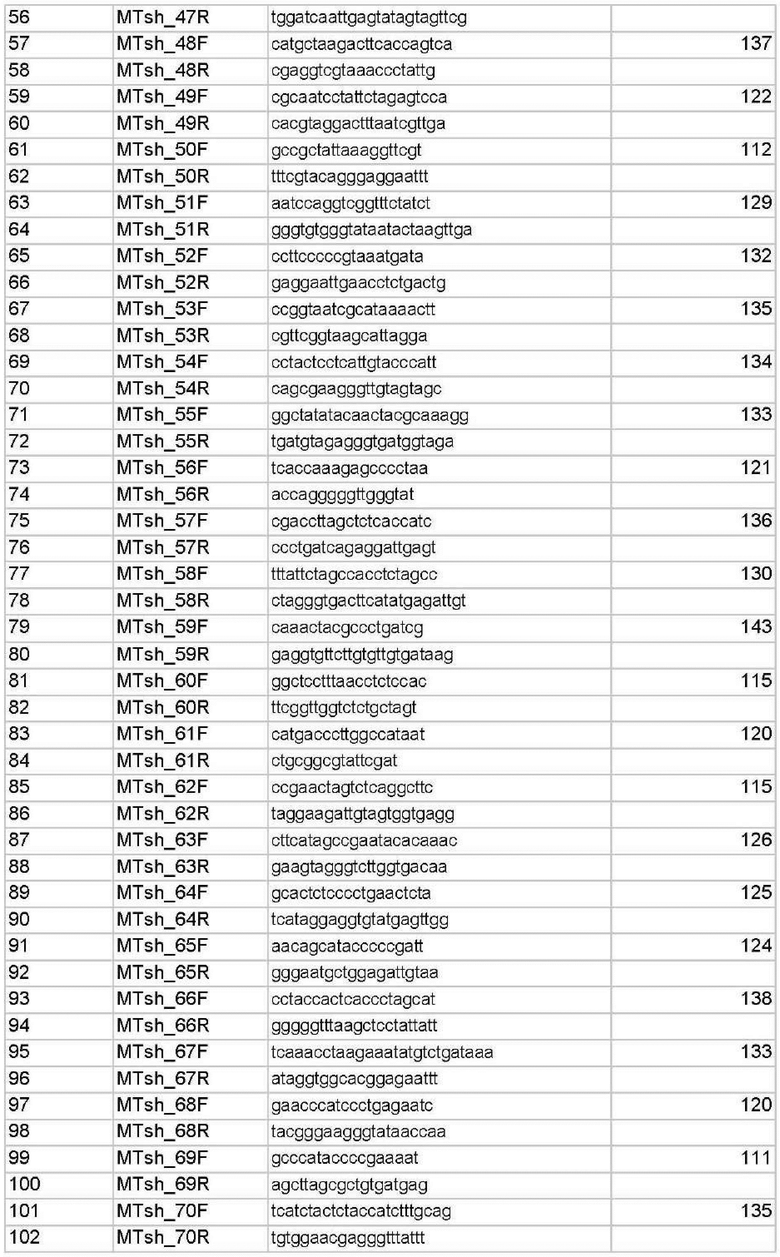
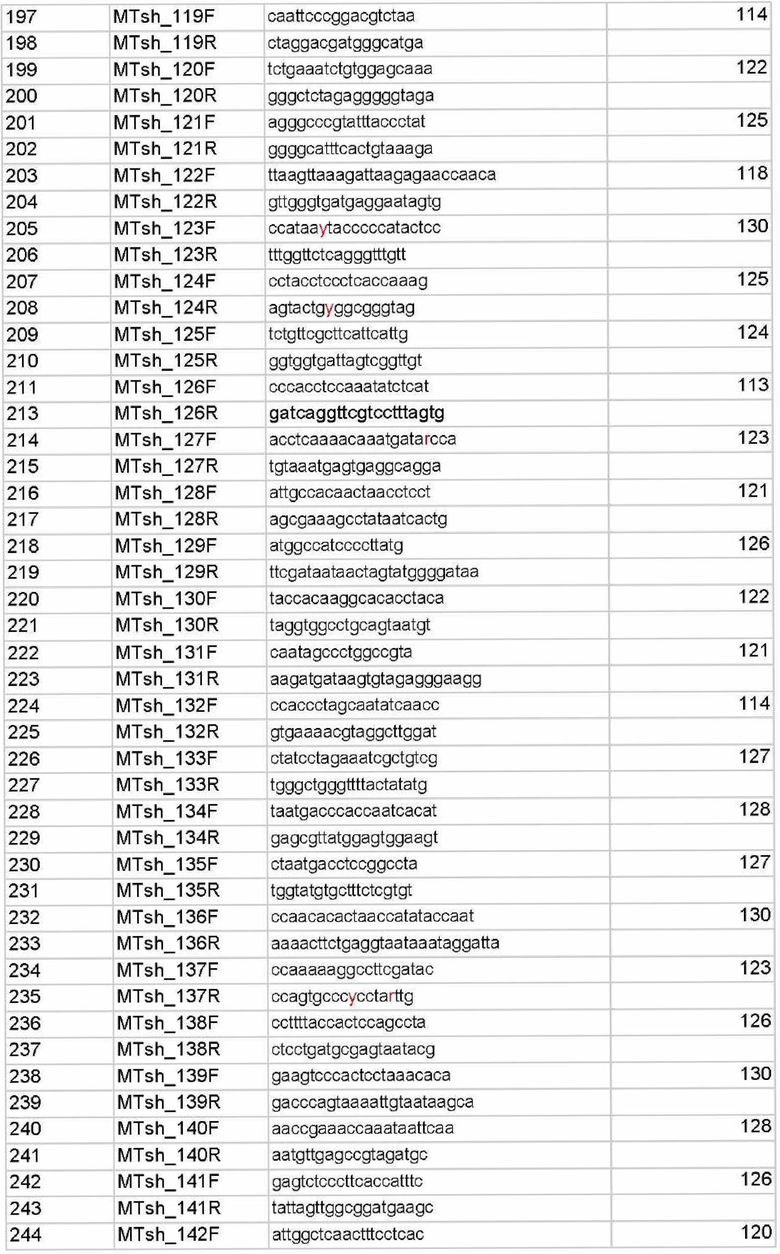
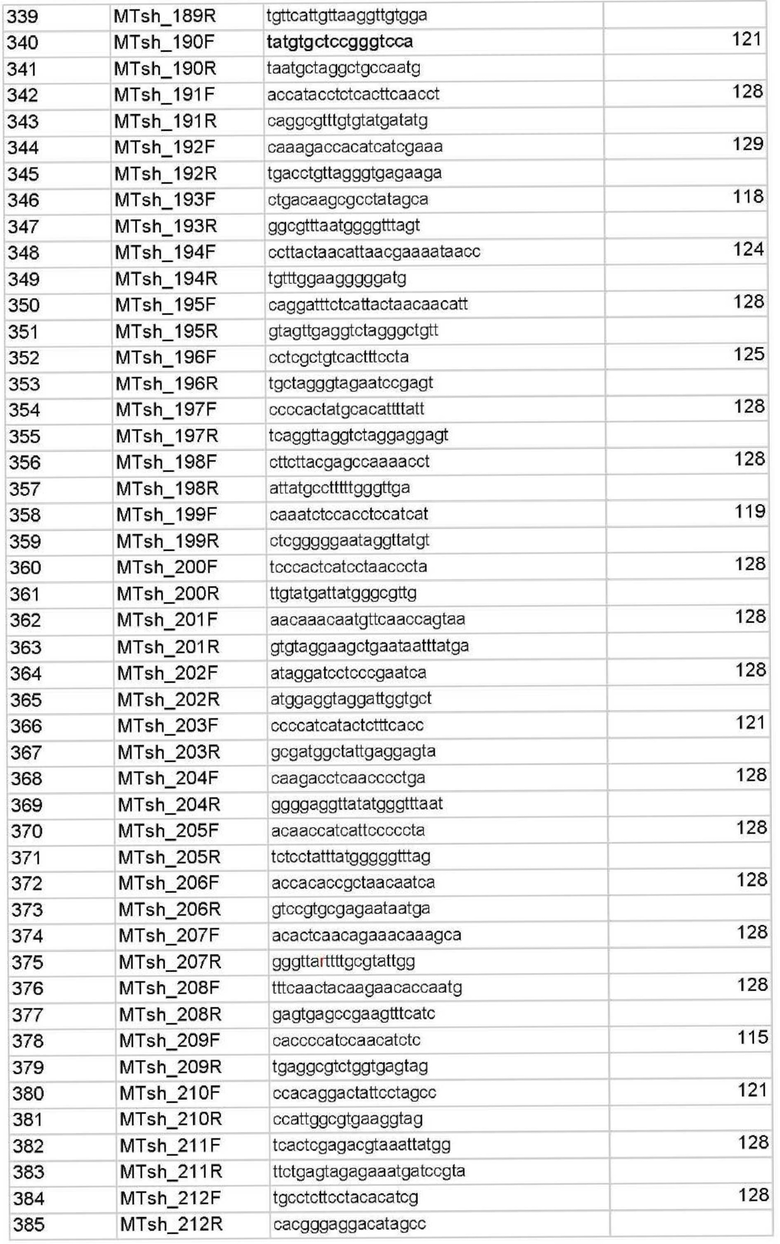
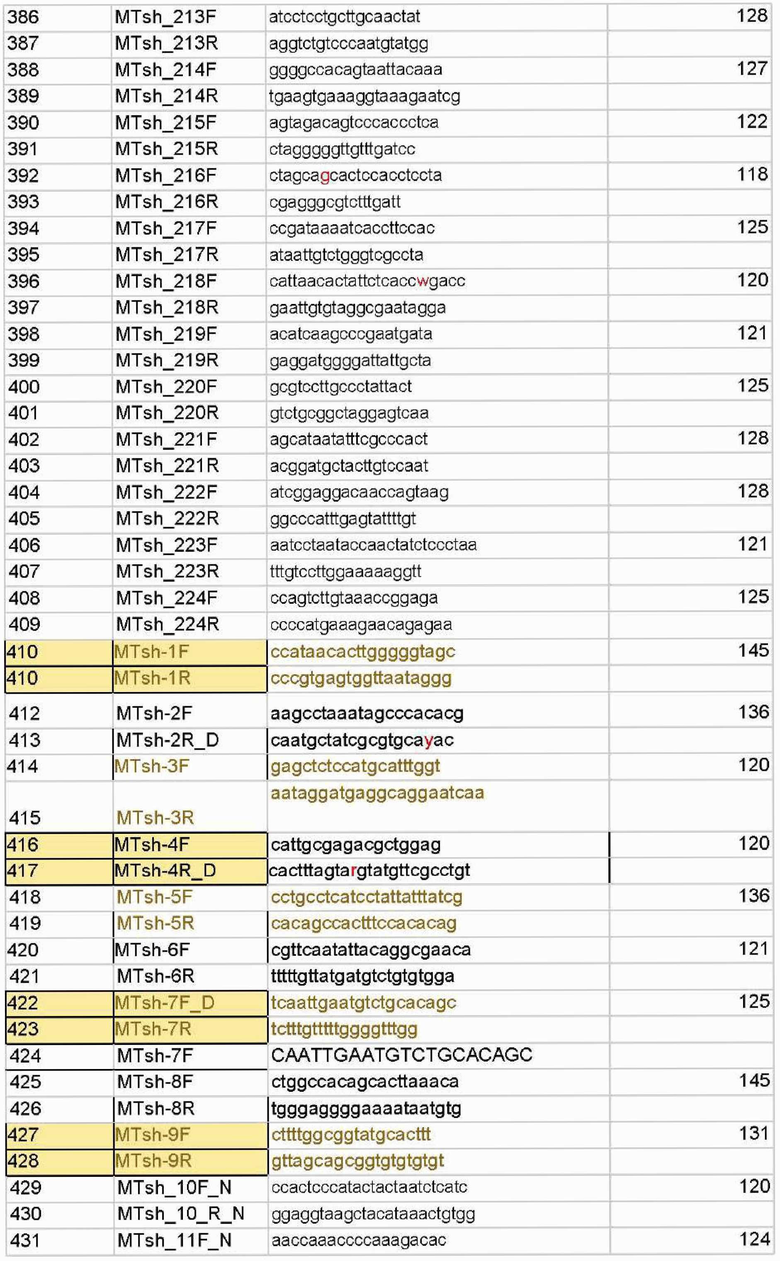
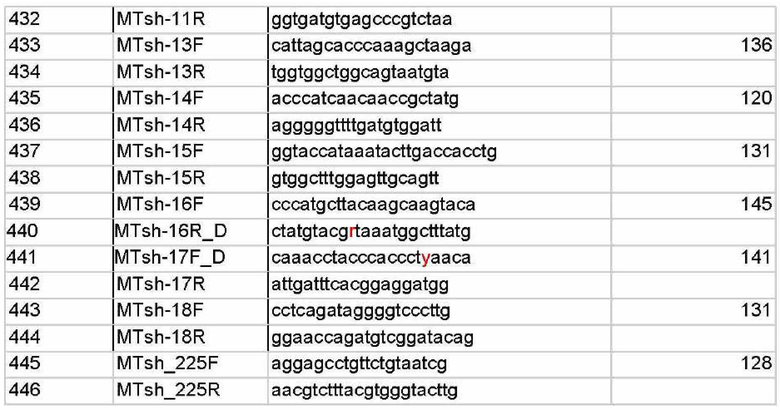
Для этих целей авторами был разработан набор из 446 олигонуклеотидных праймеров для мультиплексной ПЦР с взаимоперекрывающимися короткими ампликонами (до 145 п.о), подобранными на основе популяционных данных индивидов из российских популяций (табл.Г), позволяющий определить полную последовательности нуклеотидов митохондриальной ДНК на малых количествах ДНК и деградированной ДНК.
Таким образом, данное изобретение раскрывает разработанные синтетические оригинальные олигонуклеотидные праймеры, позволяющие амплифицировать короткие перекрывающиеся участки митохондриальной ДНК человека ддя последующего секвенирования с помощью метода «массового параллельного секвенирования» (NGS), или по методу Сенгера или другими методами секвенирования, с последующим выравниванием прочтений на референсные митохондриальные геномы индивидов из российских популяций.\
Способ предназначен:
- для получения полноразмерной последовательности митохондриальной ДНК человека в образцах ДНК, полученных из биологического материала, в котором ДНК находится в деградированном состоянии для генетической идентификации личности человека в медико-диагностических, судебно-медицинских и криминалистических лабораториях;
- для установления родственных связей, в том числе возможное происхождение индивидов по женской линии;
- для научных исследований в области популяционной генетики человека и медицинской генетики.
Изобретение позволяет проводить ДНК-идентификацию индивида по полной последовательности митохондриальной ДНК, а также определять родство и возможное происхождение индивидов по женской линии в российской популяции с высокой чувствительностью, специфичностью и точностью по сравнению с существующими аналогами. Использование изобретения позволяет проводить анализ одновременно с большим количеством биологических образцов (более 10). Преимущества данной разработки состоит в том, что авторами изобретения создана система олигонуклеотидов, учитывающая популяционный полиморфизм, встречающийся в российских популяциях, т.е. наиболее информативная система для ДНК-идентификации в популяциях России, а также предназначенная для использования на малых количествах биологического материала низкого качества.




Осуществление способа
Пример определения последовательности митохондриальной ДНК биологического образца с использованием метода «массового параллельного секвенирования» (NGS).
Данный способ описывает методику определения полной последовательности митохондриальной ДНК на образцах как полногеномной ДНК, так и деградированной ДНК с последующим "массовым параллельным секвенированием" полученных ДНК-образцов. Способ осуществляют следующим образом: для работы используют образцы геномной ДНК в том числе сильно деградированная ДНК, выделенной из любого биологического материала человека в соответствии со стандартными методами, обеспечивающими качественную очистку ДНК от белков и примесей. Определение полной последовательности мтДНК проводится на образце ДНК в количестве 100 пкг, выделенной из любого биоматериала человека.
Получение ДНК
ДНК может быть получена из любой ткани человека, например, лейкоцитов, волосяных фолликулов, клеток буккального эпителия и др. Пригодная для проведения ПЦР ДНК может быть выделена из образца при помощи любого метода выделения ДНК, который может быть в равной степени эффективным. В частности, есть несколько готовых наборов для выделения ДНК из различных тканей человека (например, представленных фирмой Qiagen)
Амплификация участков митохондриальной ДНК
Амплификация перекрывающихся участков митохондриальной ДНК может быть осуществлена с помощью наборов Multiplex PGR kit компании «Qiagen», на приборах GeneAmp PGR System 9700 Thermal Cycler компании «Applied Biosystems».
Состав реакционной смеси для мультиплексной ПНР:
-исследуемая ДНК
- полимераза (Taq-, HotTaq-, или иная);
- буфер, оптимизированный для работы соответствующей полимеразы, например: 70 mM Tris-HCl (рН 8.8 при 25°С); 17 тМ (NH4)2SO4, 0.01% Tween-20. 2 ммоль MgCl2;
- смеси дезоксирибонуклеотидтрифосфатов четырех типов (dATP, dTTP, dGTP, dCTP);
- смесь праймеров в конечной концентрации от 1 нМ до 0,5 мкМ.
Рекомендуемое количество ДНК для мультиплексной ПЦР - 0,1 нг. Также необходимо проводить параллельно контрольную реакцию (отрицательный контроль), где вместо ДНК добавляется деионизированная вода.
Амплификацию следует проводить в пробирках, стрипах, или планшетах предназначенных для ПЦР.
После приготовления реакционную смесь помещают в амплификатор и выставляется программа:

Продукты амплификации, полученные при использовании смеси олигонуклеотидов очищают от остаточных праймеров с помощью соответствующих мини-колонок (например, Qiagen MinElute system) в соответствии с инструкцией к используемому набору реагентов. Полученные в результате мультиплексной амплификации очищенные ПЦР продукты, содержащие перекрывающиеся фрагменты митохондриальной ДНК используются в качестве матрицы для приготовления фрагментных геномных библиотек для секвенирования.
Приготовление фрагментных библиотек
Предварительно необходимо провести измерение концентрации очищенных 1ЩР-фрагментов, полученных на предыдущем этапе мультиплексной амплификации на приборах NanoDrop или Qubit (ThermoFisher) в соответствии с инструкцией к прибору. Для приготовления фрагментных библиотек оптимально использовать около 300 нг ДНК очищенных ПЦР-продуктов мультиплексной амплификации. Приготовление фрагментных геномных библиотек проводят с использованием наборов реагентов Illumina®. TruSeq® DNA PCR-Free library prep kit в соответствии с инструкцией к набору. Для секвенирования нескольких образцов фрагментных библиотек одновременно необходимо использовать индексированные адаптеры (например. Dual index adaptors Illumina). Для каждого образца библиотеки используется отдельный индексируемый адаптер, который необходимо записать в таблицу для дальнейшего учета при анализе результатов секвенирования нескольких образцов фрагментных библиотек. Для приготовления фрагментных библиотек рекомендуется/возможно использовать 1/2 от объема всех реагентов, входящих в набор Illumina®. TniSeq® DNA PCR-Free library prep kit, прописанного в инструкции к набору.
Этапы 1-3 являются рекомендованными, но не обязательными.
1 Очистка фрагментных библиотек: необходимо нанести весь объем элгоата в одну лунку 2% агарозного геля с красителем SYBR Gold (например, с использованием готового 2%-ного E-gel (Invitrogen)). Провести электрофорез с использованием маркера молекулярных весов 50 bp Ladder в течение 10 минут (в случае использования системы e-gel (Invitrigen)).
2 С использованием стерильного одноразового скальпеля вырезать кусочки геля, с фрагментами библиотеки от 220 п.н. и больше. Светящиеся фрагменты длиной меньше 150 п.н. соответствуют димерам адаптеров и не должны быть использованы для дальнейшего анализа.
3 Провести очистку вырезанных фрагментов ДНК с использованием набора реагентов MinElute gele extraction kit (Qiagen). Эллюировать в 20-22 мкл буфера ЕВ (Qiagen).
Приготовленные описанным выше способом фрагментные библиотеки можно напрямую использовать для секвенирования на технологической платформе Illumina, предварительно проведя оценку качества полученных фрагментных библиотек.
Качественную и количественную оценку фрагментных библиотек можно проводить стандартными методами, рекомендованными фирмой производителем платформ для секвенирования Illumina. Для оптимизации рабочего процесса рекомендуем использовать следующие этапы: Измерить концентрации фрагментных библиотек на приборе Qubit с использованием набора реагентов HS. Для дальнейшего секвенирования рекомендуется использовать фрагментные библиотеки с концентрацией не менее 1 нМ/мкл.
Перед секвенированием необходимо провести мультиплексирование библиотек в эквимолярных количествах (объединение библиотек для нескольких образцов). Не допускается объединение в один пул библиотек с одинаковыми индексами.
Приготовленные фрагментные библиотеки секвенируют методом параллельного широкомасштабного секвенирования на технологической платформе Illumina в парноконцевом режиме с длинной прочтения не менее 75+75 п.о. Рекомендуемый прибор секвенатор MiSeq (Illumina).
Анализ результатов секвенирования
Анализ результатов секвенирования проводят следующим образом:
Картировать полученные прочтения на референсного поледовательность мтДНК человека (rCRS) с помощью программы bwamem или аналогичной.
Определить варианты митохондриальной ДНК с помощью программ GATK, freebayes или аналогичных и использовать их для реконструкции полной последовательности митохондриальной ДНК.
Литературные источники
1. Parson W. et al. DNA Commission of the International Society for Forensic Genetics: Revised and extended guidelines for mitochondrial DNA typing // Forensic Sci. Int. Genet. 2014. Vol.13. P. 134-142.
2. Kivisild T. Maternal ancestry and population history from whole mitochondrial genomes // Investig. Genet. 2015. Vol.6, №1. P. 3.
3. Andréasson H. et al. Nuclear and mitochondrial DNA quantification of various forensic materials // Forensic Sci. Int. 2006. Vol.164, №1. P. 56-64.
4. Schwartz M., Vissing J. Paternal Inheritance of Mitochondrial DNA // N. Engl. J. Med. 2002. 85 Vol.347, №8. P. 576-580.
5. Krause J. et al. Multiplex amplification of the mammoth mitochondrial genome and the evolution of Elephantidae // Nature. 2006. Vol.439. №7077. P. 724-727.
6. Chaitanya L. et al. Simultaneous Whole Mitochondrial Genome Sequencing with Short Overlapping Amplicons Suitable for Degraded DNA Using the Ion Torrent Personal Genome Machine // Hum. Mutat. 2015. Vol.36, №12. P. 1236-1247.
7. Enk J.M. et al. Ancient Whole Genome Enrichment Using Baits Built from Modem DNA // Mol. Biol. Evol. 2014. Vol.31, №5. P. 1292-1294.
8. van der Valk T. et al. Whole mitochondrial genome capture from faecal samples and museum-preserved specimens // Mol. Ecol. Resour. 2017.
9. Maricic Т., Whitten M., Pääbo S. Multiplexed DNA Sequence Capture of Mitochondrial Genomes Using PGR Products // PLoS One / ed. Fleischer R.C. 2010. Vol.5, №11. P. e 14004.
10. Hancock-Hanser B.L. et al. Targeted multiplex next-generation sequencing: advances in techniques of mitochondrial and nuclear DNA sequencing for population genomics // Mol. Ecol. Resour. 2013. Vol.13, №2. P. 254-268. 86
11. Briggs A.W. et al. Primer Extension Capture: Targeted Sequence Retrieval from Heavily Degraded DNA Sources // J. Vis. Exp.2009. №31.
12. Eduardoff M. et al. Optimized mtDNA Control Region Primer Extension Capture Analysis for Forensically Relevant Samples and Highly Compromised mtDNA of Different Age and Origin // Genes (Basel). 2017. Vol. 8, №10. P. 237.










Изобретение относится к области молекулярной биологии. Описан способ получения полноразмерной последовательности митохондриальной ДНК человека для идентификации неизвестного индивида и определения биологического родства методом мультиплексной амплификации для работы с образцами деградированной ДНК. Способ характеризуется тем, что для генотипирования митохондриальной ДНК используется композиция из 446 оригинальных синтетических олигонуклеотидных праймеров для проведения мультиплексной полимеразной цепной реакции, фланкирующих участки ДНК, содержащие короткие перекрывающиеся участки митохондриальной ДНК человека. Технический результат заключается в подборе синтетических праймеров, позволяющих амплифицировать короткие перекрывающиеся участки митохондриальной ДНК человека для последующего секвенирования с помощью метода «массового параллельного секвенирования» (NGS), или по методу Сенгера, или другими методами секвенирования, с последующим выравниванием прочтений на референсные митохондриальные геномы индивидов из российских популяций. 2 н. и 3 з.п. ф-лы, 2 табл., 1 пр.
1. Способ получения полноразмерной последовательности митохондриальной ДНК человека для идентификации неизвестного индивида и определения биологического родства методом мультиплексной амплификации для работы с образцами деградированной ДНК, характеризующийся тем, что для генотипирования митохондриальной ДНК используется композиция из 446 оригинальных синтетических олигонуклеотидных праймеров для проведения мультиплексной полимеразной цепной реакции (ПЦР) с нуклеотидными последовательностями SEQ ID NO: 1-SEQ ID NO 446, фланкирующих участки ДНК, содержащие короткие перекрывающиеся участки митохондриальной ДНК человека; ПЦР-продукты, соответствующие выбранным праймерам, представляют собой маркеры.
2. Способ по п.1, отличающийся тем, что размер полученных ПЦР-продуктов составляет не более 145 п.н.
3. Способ по п.1, отличающийся тем, что полученные в результате мультиплексной амплификации ПЦР продукты, содержащие короткие перекрывающиеся участки митохондриальной ДНК человека, используются в качестве матрицы для различных методов генетического определения нуклеотидной последовательности.
4. Способ по п.1, отличающийся тем, что определение нуклеотидной последовательности продуктов амплификации, ПЦР-продуктов, проводят секвенированием по методу массового параллельного секвенирования (NGS), секвенированием по методу Сэнгера, методом ПЦР в реальном времени с использованием флуоресцентных зондов типа TaqMan, а также проводят любым доступным способом определения нуклеотидной последовательности.
5. Набор олигонуклеотидов для осуществления способа по по п.1, используемый для получения полноразмерной последовательности митохондриальной ДНК человека, включающий синтетические оригинальные праймеры с нуклеотидными последовательностями SEQ ID NO: 1-SEQ ID NO: 446.
| Timmermans M | |||
| J | |||
| T | |||
| N | |||
| et al | |||
| Why barcode? High-throughput multiplex sequencing of mitochondrial genomes for molecular systematics //Nucleic acids research | |||
| Приспособление для суммирования отрезков прямых линий | 1923 |
|
SU2010A1 |
| - Т | |||
| Способ сужения чугунных изделий | 1922 |
|
SU38A1 |
| - No | |||
| Выбрасывающий ячеистый аппарат для рядовых сеялок | 1922 |
|
SU21A1 |
| - С | |||
| Способ утилизации отработанного щелока из бучильных котлов отбельных фабрик | 1923 |
|
SU197A1 |
| Newell C | |||
| et al | |||
| Plasma-derived cell-free mitochondrial DNA: A novel non-invasive methodology to identify mitochondrial DNA haplogroups in humans | |||
Авторы
Даты
2024-05-02—Публикация
2021-11-10—Подача