Содержание поданного в электронном виде списка последовательностей в текстовом ASCII-файле (название: 4349.001PC01_Seqlisting_ST25; размер: 389 120 байт; дата создания: 11 июня 2020 г.), поданном вместе с заявкой, включено сюда путем ссылки во всей полноте.
Уровень техники
Общим для многих заболеваний типа рака, пороков развития и некоторых инфекций являются генетические и эпигенетические аберрации. Генная терапия предназначена для введения генетического материала в клетки для нацеливания и непосредственного редактирования генома затем, чтобы исправить генетически нарушенные клетки и тем самым вылечить связанные с ними заболевания. Цинк-пальцевые нуклеазы (ZFN), технологии редактирования генов Talen и CRISPR-Cas9 представляют некоторые из недавно разработанных инструментов для редактирования ДНК. Для доставки генетического материала в геном применялись такие методы, как электропорация, катионные липиды, микроинъекции или вирусы. Современные стратегии доставки генов обычно основываются на аденовирусах, ретровирусах или “голых” ДНК-плазмидах.
Лентивирусы, в том числе ВИЧ, являются мощным инструментом при использовании в качестве вектора для доставки нуклеиновых кислот. Лентивирусы способны стабильно инфицировать делящиеся и неделящиеся клетки. Лентивирусные векторы склонны к случайной интеграции в геном хозяина и часто могут встраиваться на участке сильно транскрибируемых генов, что повышает риск вставочного мутагенеза.
Интеграза ВИЧ-1 катализирует встраивание вирусной ДНК в геном хозяина. В общем, интеграза ВИЧ-1 состоит из N-концевого домена (NTD), каталитического центрального домена (CCD) и C-концевого домена (CTD). NTD используется для связывания и координации катиона Zn2+ в качестве важного кофактора, а CTD используется для связывания ДНК. CCD образует каталитическое ядро, в котором катализируется процесс встраивания. Проблемы с механизмами встраивания у вирусных векторов включают низкую эффективность и отсутствие специфичности, что может привести к непреднамеренному вставочному мутагенезу и генотоксичности.
Сущность изобретения
В некоторых аспектах настоящего изобретения предусмотрены конструкции, плазмиды, векторы, частицы, слитые белки, композиции, способы и наборы, которые применимы для направленного редактирования нуклеиновых кислот, включая редактирование отдельного сайта или участка в геноме субъекта, например, геноме человека.
В приведенных здесь рабочих примерах представлены подробные экспериментальные данные, убедительно свидетельствующие об успешном создании конструкций для слитых белков программируемых транспозаз и интеграз с белками Cas9/цинковый палец. Кроме того, такие конструкции способны вызывать сайт-специфичное встраивание экзогенной последовательности нуклеиновой кислоты в геном трансфицированных клеток. Не придерживаясь какой-либо теории, авторы настоящего изобретения считают, что это первый случай, когда были созданы слитые белки такого типа, обладающие способностью к сайт-специфичному встраиванию экзогенной нуклеиновой кислоты в геном и подходящие для генной терапии, особенно с участием крупных генов. Авторы изобретения также идентифицировали модифицированные гиперактивные транспозазы PiggyBac, которые осуществляют специфичные направленные транспозиции.
Соответственно, в одном аспекте настоящего изобретения предусмотрены конструкции из нуклеиновой кислоты, включающие:
a) последовательность первого полинуклеотида, содержащего нуклеиновую кислоту, кодирующую первый ДНК-связывающий белок, сконструированный для связывания с определенной последовательностью геномной ДНК в геноме; причем первый ДНК-связывающий белок представляет собой цинк-пальцевый белок или белок Cas9;
b) вторую последовательность полинуклеотида, содержащего нуклеиновую кислоту, кодирующую второй ДНК-связывающий белок, который обеспечивает встраивание экзогенной нуклеиновой кислоты в геном, причем второй ДНК-связывающий белок представляет собой:
i. гиперактивную транспозазу PiggyBac или модифицированную гиперактивную PiggyBac с улучшенной специфичностью встраивания экзогенной нуклеиновой кислоты в геном по сравнению с гиперактивной PiggyBac либо
ii. интегразу вируса иммунодефицита человека (ВИЧ) или модифицированную интегразу ВИЧ с улучшенной специфичностью встраивания экзогенной нуклеиновой кислоты в геном по сравнению с интегразой ВИЧ; и
c) необязательно последовательность полинуклеотида, содержащего нуклеиновую кислоту, кодирующую линкер;
причем конструкция нуклеиновой кислоты кодирует слитый белок, включающий первый ДНК-связывающий белок, второй ДНК-связывающий белок и необязательно линкер между первым ДНК-связывающим белком и вторым ДНК-связывающим белком; и
при этом слитый белок обеспечивает встраивание экзогенной нуклеиновой кислоты в определенное место генома.
Также предусмотрены композиции, включающие конструкцию из нуклеиновой кислоты, вектор или слитый белок, как описано здесь, и последовательность полинуклеотида, кодирующего экзогенную нуклеиновую кислоту для встраивания в геном, причем композиции содержатся в упаковочном векторе или связаны с ним.
Настоящим изобретением также предусмотрен способ контролируемого сайт-специфичного встраивания одной копии или нескольких копий экзогенной последовательности нуклеиновой кислоты в клетки, включающий: (a) введение в клетки конструкции из нуклеиновой кислоты, вектора или слитого белка, описанных здесь, и (b) введение в клетки экзогенной нуклеиновой кислоты; причем связывание слитого белка с определенной последовательностью геномной ДНК в геноме клеток приводит к расщеплению генома и встраиванию одной или нескольких копий экзогенной нуклеиновой кислоты в геном клеток.
В другом аспекте предусмотрено получение модифицированных гиперактивных транспозаз PiggyBac, включающих аминокислотную последовательность SEQ ID NO: 9, где: в положении 245 находится аминокислота A, в положении 275 – аминокислота R или A, в положении 277 – аминокислота R или A, в положении 325 – аминокислота A или G, в положении 347 – аминокислота N или A, в положении 351 – аминокислота E, P или A, в положении 372 – аминокислота R, в положении 375 – аминокислота A, в положении 450 – аминокислота D или N, в положении 465 – аминокислота W или A, в положении 560 – аминокислота T или A, в положении 564 – аминокислота P или S, в положении 573 – аминокислота S или A, в положении 592 – аминокислота G или S, а в положении 594 – аминокислота L или F.
В некоторых воплощениях предусмотрены слитые белки (i) интегразы, модифицированной интегразы, транспозазы или модифицированной транспозазы, слитые с (ii) белком Cas9 или цинк-пальцевым белком; и кодирующие их конструкции из нуклеиновой кислоты.
В некоторых аспектах изобретения предусмотрены конструкции из нуклеиновой кислоты, включающие: (a) последовательность первого полинуклеотида, кодирующего первый ДНК-связывающий белок, сконструированный для связывания с определенной последовательностью геномной ДНК в геноме; (b) последовательность второго полинуклеотида, кодирующего второй ДНК-связывающий белок, который обеспечивает встраивание экзогенной нуклеиновой кислоты в геном, причем второй ДНК-связывающий белок представляет собой (i) интегразу или модифицированную интегразу, которая модифицирована относительно интегразы дикого типа, либо (ii) транспозазу или модифицированную транспозазу, которая модифицирована относительно транспозазы дикого типа; и (c) последовательность третьего полинуклеотида, содержащего нуклеиновую кислоту, кодирующую линкер; причем конструкции из нуклеиновой кислоты кодируют слитые белки, включающие первый ДНК-связывающий белок, второй ДНК-связывающий белок и линкер между первым ДНК-связывающим белком и вторым ДНК-связывающим белком.
В некоторых воплощениях конструкции из нуклеиновой кислоты включают: (a) последовательность первого полинуклеотида, кодирующего белок Cas9; и (b) последовательность второго полинуклеотида, кодирующего транспозазу или модифицированную гиперактивную транспозазу PiggyBac по изобретению либо её функциональный фрагмент.
В некоторых воплощениях конструкции из нуклеиновой кислоты включают: (a) последовательность первого полинуклеотида, кодирующего цинк-пальцевый белок; и (b) последовательность второго полинуклеотида, кодирующего интегразу или модифицированную интегразу по изобретению либо её функциональный фрагмент.
В некоторых воплощениях изобретения предусмотрены плазмиды, векторы или клетки хозяина, содержащие конструкции из нуклеиновой кислоты по изобретению.
В некоторых аспектах изобретения предусмотрены слитые белки, включающие: первый ДНК-связывающий белок, сконструированный для связывания с определенной последовательностью геномной ДНК в геноме; второй ДНК-связывающий белок, который обеспечивает встраивание экзогенной нуклеиновой кислоты в геном, причем второй ДНК-связывающий белок представляет собой интегразу, транспозазу либо модифицированную интегразу или транспозазу; и линкер, соединяющий первый белок и второй белок.
В некоторых воплощениях слитый белок включает: (a) белок Cas9; и (b) гиперактивную транспозазу PiggyBac или модифицированную гиперактивную PiggyBac по изобретению либо её функциональный фрагмент.
В некоторых воплощениях слитый белок включает: (a) цинк-пальцевый белок; и (b) интегразу или модифицированную интегразу по изобретению либо её функциональный фрагмент.
В некоторых аспектах изобретения предусмотрены лентивирусные частицы, содержащие слитые белки по изобретению.
В некоторых аспектах изобретения предусмотрен способ встраивания последовательности экзогенной нуклеиновой кислоты в геномную ДНК организма, включающий: введение в организм лентивирусных частиц, содержащих конструкцию из нуклеиновой кислоты или слитый белок по изобретению с тем, чтобы первый и второй ДНК-связывающие белки связались с определенной последовательностью геномной ДНК и вставили экзогенную нуклеиновую кислоту в геномную ДНК; причем экзогенная нуклеиновая кислота встраивается в определенную последовательность геномной ДНК.
В некоторых аспектах изобретения предусмотрен способ контролируемого сайт-специфичного встраивания одной копии или нескольких копий экзогенной последовательности нуклеиновой кислоты в клетки, включающий: (a) введение в клетки слитого белка по изобретению и (b) введение в клетки экзогенной нуклеиновой кислоты; причем связывание слитого белка с определенной последовательностью геномной ДНК в геноме клеток приводит к расщеплению генома и встраиванию одной или нескольких копий экзогенной нуклеиновой кислоты в геном клеток; при этом слитый белок вводится в клетки при помощи лентивирусных частиц.
По всему описанию и формуле изобретения слово “включает” и его варианты не должны исключать другие технические характеристики, добавки, компоненты или стадии. Другие цели, преимущества и особенности изобретения станут очевидными специалистам в данной области после изучения описания или могут быть изучены при осуществлении изобретения на практике. Кроме того, настоящее изобретение охватывает все возможные комбинации описанных здесь конкретных и предпочтительных воплощений. Следующие ниже примеры и чертежи представлены здесь в целях иллюстрации и не должны ограничивать настоящее изобретение.
Краткое описание фигур
На фиг. 1A и 1B представлен процент клеток, содержащих экзогенную последовательность нуклеиновой кислоты, встроенную в их геном после трансфекции с помощью (фиг. 1A) слитых белков Cas9-PiggyBac (фиг. 1A) (Cas9 человека (hCas9), никазой Cas9 (nCas9) или «мертвым» Cas9 (dCas9) и гиперактивной транспозазой PiggyBac (PB)) и (фиг. 1B) слитыми белками Cas9-SB100 (Cas9 человека (hCas9), никазой Cas9 (nCas9) или «мертвым» Cas9 (dCas9) и гиперактивной транспозазой Sleeping Beauty (SB100)). Создавали векторы, в которых 3′-конец Cas9 соединяется с 5′-концом каждой из транспозаз при помощи линкера GGS (SEQ ID NO: 48, 49) (hCas9PB, nCas9PB, dCas9PB, hCas9SB, nCas9SB и dCas9SB). Создавали и другие векторы, в которых 3′-конец каждой из транспозаз соединяется с 5′-концом Cas9 при помощи линкера GGS (SEQ ID NO: 48, 49) (PBhCas9, PBnCas9, PBdCas9, SBhCas9, SbnCas9 и SBdCas9). В качестве положительного контроля использовали “PiggyBac” (фиг. 1A) и “SB100” (фиг. 1B), а в качестве отрицательных контролей – одни транспозоны, кодирующие RFP (обозначен как “эписомальный RFP” на фиг. 1A) и GFP (обозначен как “ эписомальный GFP”) на фиг.1B). На фиг. 1C приведено другое представление фиг. 1A, представляющее активность транспозиции с PB и Cas9 в различных конфигурациях.
На фиг. 2A представлена конструкция плазмиды, кодирующей слитый белок Cas9/PB.
На фиг. 2B представлен процент клеток, содержащих экзогенную последовательность нуклеиновой кислоты, встроенную в их геном при помощи слитых конструкций, образованных Cas9 человека-PiggyBac (“прицельная hCas9”) или никазой Cas9-PiggyBac (“прицельная nCas9”). 3′-конец Cas9 соединяется с 5′-концом транспозазы при помощи линкера. “Неприцельно” – это контроль на введение вообще (только PiggyBac), а “эписомально” – отрицательный контроль на отсутствие встраивания (только транспозон).
На фиг. 3 представлен типичный слитый белок ZFP-интеграза. ZFP и интеграза соединяются последовательностью GGS. NLS означает последовательность ядерной локализации.
На фиг. 4 представлены титры лентивируса: лентивируса для интегразы дикого типа (LV), пустых вирусных частиц (LVO), невстраивающегося лентивируса (NILV), невстраивающегося лентивируса с интегразой дикого типа (NILV + IN), невстраивающегося лентивируса со слитым белком ZFP-интеграза (NILV + ZP-IN (AAVS1)), невстраивающегося лентивируса со слитым белком Cas9-интеграза (NILV + Cas-IN) и лентивируса дикого типа для интегразы с интегразой дикого типа (LV + IN). ( ' ) означает технический повтор.
На фиг. 5 представлен процент клеток со встроившейся (полное встраивание) в их геном экзогенной последовательностью нуклеиновой кислоты после трансфекции лентивирусом для интегразы дикого типа (LV), пустыми вирусными частицами (LVO), невстраивающимся лентивирусом (NILV), невстраивающимся лентивирусом с интегразой дикого типа (NILV + IN), невстраивающимся лентивирусом со слитым белком ZFP-интеграза (NILV + ZP-IN (AAVS1)), невстраивающимся лентивирусом со слитым белком Cas9-интеграза (NILV + Cas-IN) и лентивирусом для интегразы дикого типа с интегразой дикого типа (LV + IN). Для каждого условия, слева направо: первый столбец – день 3, второй столбец – день 5, третий столбец – день 7, четвертый столбец – день 10 и пятый столбец – день 12.
На фиг. 6 представлено изображение хромосом с репрезентативными сайтами встраивания и не встраивания AAVS1. Звездочкой обозначен сайт для AAVS1 на хромосоме 19, треугольниками – сайты нецелевой вставки, а ромбиками – сайты направленной вставки.
На фиг. 7A представлены титры вируса: лентивируса для интегразы дикого типа (LV), пустых вирусных частиц (LVO), невстраивающегося лентивируса (NILV), невстраивающегося лентивируса с интегразой дикого типа (NILV + IN), невстраивающегося лентивируса со слитым белком ZFP-IN, нацеленным на сайт AAVS1 (NILV + ZP-IN (AAVS1)), и невстраивающегося лентивируса со слитым белком ZFP-IN, нацеленным на сайт CCR5 (NILV + ZP-IN (CCR5)).
На фиг. 7B представлен процент клеток со встроившейся (полное встраивание) в их геном экзогенной последовательностью нуклеиновой кислоты после трансфекции лентивирусом для интегразы дикого типа (LV), невстраивающимся лентивирусом (NILV), невстраивающимся лентивирусом с интегразой дикого типа (NILV + IN), невстраивающимся лентивирусом со слитым белком ZFP-IN, нацеленным на сайт AAVS1 (NILV + ZP-IN (AAVS1)), и невстраивающимся лентивирусом со слитым белком ZFP-IN, нацеленным на сайт CCR5 (NILV + ZP-IN (CCR5)).
На фиг. 7C представлен процент клеток со встроившейся (полное встраивание) в их геном экзогенной последовательностью нуклеиновой кислоты после трансфекции лентивирусом для интегразы дикого типа (LV), пустыми вирусными частицами (LVO), невстраивающимся лентивирусом (NILV), невстраивающимся лентивирусом с интегразой дикого типа (NILV + IN), невстраивающимся лентивирусом со слитым белком ZFP-IN, нацеленным на сайт AAVS1 (NILV + ZP-IN (AAVS1)), и невстраивающимся лентивирусом со слитым белком ZFP-IN, нацеленным на сайт CCR5 (NILV + ZP-IN (CCR5)).
На фиг. 7D представлен процент клеток со встроившейся (полное встраивание) в их геном экзогенной последовательностью нуклеиновой кислоты после трансфекции лентивирусом для интегразы дикого типа (LV), невстраивающимся лентивирусом (NILV), невстраивающимся лентивирусом с интегразой дикого типа (NILV + IN), невстраивающимся лентивирусом со слитым белком ZFP-IN, нацеленным на сайт AAVS1 (NILV + ZP-IN (AAVS1)), и невстраивающимся лентивирусом со слитым белком ZFP-IN, нацеленным на сайт CCR5 (NILV + ZP-IN (CCR5)).
На фиг. 8A-8C представлены титры лентивируса (фиг. 8A) и % экспрессирующих CAR клеток в день 3 и в день 14 (фиг. 8B), а на фиг. 8C представлен % экспрессирующих CD3 клеток. Клетки Jurkat трансфицировали лентивирусом в нескольких состояниях: лентивирусом для интегразы дикого типа (LV), пустыми вирусными частицами (LVO), невстраивающимся лентивирусом (NILV), невстраивающимся лентивирусом с интегразой дикого типа (NILV + IN), невстраивающимся лентивирусом со слитым белком ZFP-интеграза (NILV + ZFP-IN (TRCa-1)), и невстраивающимся лентивирусом со слитым белком Cas9-интеграза (NILV + Cas-IN). NILV проявлял резкое снижение титра, а транскомплементация с экспрессией IN дикого типа или слитого белка ZNF-IN в вырабатывающих вирус клетках не давала восстановления ни титра, ни способности к встраиванию. Кроме того, клетки не теряли экспрессию CD3, когда встраивание было нацелено на локус TCR (экспрессия белка CD3). Это значит, что нужно использовать дополнительные факторы для транскомплементации типа белка VPR, особенно в контексте этой линии клеток.
На фиг. 9A-9B представлены титры лентивируса WT и двух различных вирусных систем, дефектных по интегразе (NILV и TAA, последнее означает то, что был введен стоп-кодон в начало области, кодирующей IN в упаковочной плазмиде для лентивируса), отдельно или с транскомплементацией IN или слитым белком VPR_IN. Титры определяли методом флуоресцентной цитометрии в день 3 после трансфекции (фиг. 9A). На фиг. 9B представлена относительная эффективность встраивания по механизмам встраивания с транскомплементацией, показывающая преимущество слитого с VPR белка перед IN для транскомплементации. WT: лентивирус, полученный с IN дикого типа; NILV: лентивирус, полученный с невстраивающейся IN, несущей две мутации в своем каталитическом центре; TAA: лентивирус, полученный с дефектной IN, у которой не экспрессируется белок; +IN: лентивирус с транскомплементацией IN; +VPR-IN: лентивирус с транскомплементацией IN, слитой с VPR на С-конце.
На фиг. 10A представлена схема конструкции из нуклеиновой кислоты, образованной из домена встраивания с ДНК-связывающим доменом и программируемого домена распознавания ДНК, слитых при помощи линкера. На фиг. 10B представлена схема слияния Cas9 и транспозазы, соединенных линкером в различных конфигурациях.
На фиг. 11 представлены результаты по активности Cas9 у Cas9, связанной с hyPB с помощью линкеров различного размера и состава. Активность Cas9 измеряли путем секвенирования сайта мишени направляющей РНК и анализа частоты инделей с помощью CRISPR-GA. Использовали 2 разные направляющие РНК, нацеленные на сайт AAVS1. Использовали линкеры по SEQ ID NO: 50-63.
На фиг. 12 представлены результаты по эффективности транспозиции программируемой транспозазы Genetrap. Флуоресценцию RFP измеряли методом проточной цитометрии через 10 дней после трансфекции. Использовали различные линкеры, чтобы определить важность длины и состава линкеров для направленного встраивания. Средние значения из 2 независимых экспериментов. Использовали линкеры по SEQ ID NO: 50-63.
На фиг. 13 представлены результаты по направленной транспозиции с линкерами hcas9_PB. Эффективность направленной транспозиции у конструкций с различными линкерами cas9-PB измеряли на клетках линии с расщепленным GFP, используя 2 разные направляющие РНК. Экспрессию GFP измеряли методом проточной цитометрии через 72 ч после трансфекции.
На фиг. 14 представлена схема репортерной линии клеток с расщепленным GFP, созданной для скрининга и высокопроизводительного анализа библиотек различных мутаций hyPB, а также для проверки отдельных мутантов. В геном клеток HEK293T вводили акцептор сплайсинга (SA) вместе с половиной кодирующей последовательности GFP (Ct-GFP) после сайта мишени с помощью системы Sleeping Beauty 100x. Для такого скрининга транспозон PiggyBac, фланкированный инвертированными концевыми повторами (ITR), представлял собой либо полную кассету для экспрессии RPF вместе с промотором, другой половиной GFP (Nt-GFP) и донором сплайсинга (SD); либо только половинку GFP; как показано на рисунке.
На фиг. 15 представлены результаты по направленной транспозиции выбранных мутантов hCas9_PB. Эффективность направленной транспозиции hCas9_PB D450N и hCas9_PB R372A K375A D450. Измеряли экспрессию GFP методом проточной цитометрии через 72 ч после трансфекции. Средние значения из 4 независимых экспериментов.
На фиг. 16 представлены результаты по случайной и направленной транспозиции выбранных мутантов hCas9_PB. Эффективность случайной и прицельной транспозиции hCas9_PB D450N и hCas9_PB R372A K375A D450. Измеряли экспрессию GFP методом проточной цитометрии через 72 ч после трансфекции и экспрессию RFP методом проточной цитометрии через 15 дней после трансфекции и нормировали по флуоресценции RFP через 48 ч после трансфекции, принимая в качестве эффективности трансфекции.
На фиг. 17 представлена схема слияния ZFP и транспозазы, соединенных линкером в различных конфигурациях.
На фиг. 18 представлены результаты по направленной транспозиции слитых белков ZFP-PB. Эффективность направленной транспозиции ZFP_hyPB и ZFP_hyPBD450N в N- и C-концевой конформации. Измеряли экспрессию GFP методом проточной цитометрии через 5 дней после трансфекции. Более 1 независимого повтора. ZFP_PB: слияние ZFP и hyPB в C-концевой конфигурации с помощью линкера XTEN; PB_ZFP: слияние ZFP и hyPB в N-концевой конфигурации с помощью линкера XTEN, ZFP_450: слияние ZFP и hyPB (D450N) в C-концевой конфигурации с помощью линкера XTEN; 450_ZFP: слияние ZFP и hyPB (D450N) в N-концевой конфигурации с помощью линкера XTEN; hyPB: hyPB без модификаций; 1/2 GFP: только контрольный транспозон.
На фиг. 19 представлена схема метода анализа, используемого при скрининге библиотеки мутаций PiggyBac.
На фиг. 20 представлено секвенирование области PiggyBac в 1116 п.н. у всех вариантов из библиотеки по технологии Illumina NGS. Праймер Index I7 заменяли на специальный праймер для полного секвенирования различных вариантов, за исключением вариантов 450 и 465.
На фиг. 21A-21B представлены результаты по созданию библиотеки разнообразия hyPB. На фиг. 21A представлен пример графика сортировки. Положительные результаты направленного встраивания (флуоресценция GFP) отбирали в канале P4, а отрицательные результаты направленного встраивания (отсутствие флуоресценции GFP) отбирали в канале P5. Нежизнеспособные клетки и обломки проходили как отрицательные в других каналах по окрашиванию DAPI. На фиг. 21B представлены результаты по эффективности трансфекции двумя плазмидами. Эффективность трансфекции измеряли при трансфекции плазмидами с GFP и RFP эквимолярно по ½ GFP и трансфекции направляющей РНК в один и тот же день и в тех же условиях. Трансфекция двумя плазмидами проходит по каналу P8. Нежизнеспособные клетки и обломки проходили как отрицательные в других каналах по окрашиванию DAPI.
На фиг. 22A-22K представлены результаты анализа скрининга библиотеки при сравнении положительных результатов с отрицательными. Фиг. 22A-22B: секвенирование массива библиотеки в качестве контроля качества; при этом подавляющее большинство вариантов представлено только один раз. Представлена логограмма массива репрезентативной библиотеки PiggyBac, в которой положения соответствуют положениям аминокислот: 1 - R245; 2 - R275; 3 - R277; 4 - G325; 5 - N347; 6 - S351; 7 - R372; 8 - K375; 9 - R388; 10 - T560; 11 - S564; 12 - S573; 13 - M589; 14 - S592; 15 - F594. Кроме того, представлена логограмма для прошедших отрицательный отбор клеток с таким же профилем, что и для массива библиотеки. Фиг. 22C-22K соответствуют 3 независимым повторам положительных попаданий; варианты для положительных логограмм (внизу), а также самый лучший вариант Лучший 1 после отбора (вверху). Также представлены логограммы для 5 лучших и 10 лучших вариантов. На левых панелях B, C представлено относительное обогащение вариантов PiggyBac в прошедших положительный и отрицательный отбор популяциях по шкале log2.
На фиг. 23A представлены положительные варианты Лучший 1 и Лучший 3 из независимого повтора 3. Имеется отличие только по 1 аминокислоте в положении 254. На фиг. 23B представлены 3 варианта Лучший 1, идентифицированные в 3 независимых повторах. Также представлен hyPB дикого типа для сравнения.
На фиг. 24A представлены наиболее распространенные варианты в GFP-положительных клетках в сравнении с RFP-положительными клетками. Представлена кластеризация: GPF, направленное встраивание; RPF, случайное встраивание; и отрицательная популяция. На фиг. 24B и 24C представлены варианты, обнаруженные среди положительных попаданий более чем в 1 независимом повторе. Rep: независимый повтор эксперимента; Pos: положительные клетки с направленной вставкой; Neg: отрицательные клетки, в которых не произошло направленного встраивания.
На фиг. 25 представлена гистограмма ковариации вариантов. Представлены проценты, когда один вариант отмечен вместе с другим в положительных образцах, деленные на отрицательные образцы. Наряду с вариантами, включенными при разработке библиотеки, анализировали и варианты со случайными вставками, введенными под действием обратной транскриптазы лентивируса при получении вирусной библиотеки. Некоторые из этих новых вариантов связаны с положительными попаданиями и выполняют направленное встраивание при комбинировании. Примеры – D450N и W465A.
На фиг. 26 показано, что модифицированная hyPB проявляет большее повышение направленного встраивания, чем hyPB дикого типа при слиянии с Cas9. Проводили слияние Cas9 с hyPB или с различными комбинациями мутантов hyPB (Unilarge-A: D450N; Unilarge-B: R245A/D450N; Unilarge-C: R245A/G325A/D450N/S573P; Unilarge-D: R245A/G325A/S573P), используя линкер 4GGS и систему репортерных линий клеток.
На фиг. 27 представлены результаты по транскомплементации с дефектной интегразой. Определяли эффективность продукции вируса при измерении на 2-й день и способность к встраиванию при измерении на 7-й день для различных систем в клетках HEK293T. Вестерн-блоттинг показал наличие IN в вирусных частицах in trans. Эффективность продукции вируса и его способность к встраиванию определяли путем трансфекции дефектного по встраиванию вируса и транскомплементированного вируса в HEK293T при различных условиях. Клетки пассировали в течение 7 дней до тех пор, пока не проявлялось отсутствие эписомального сигнала, и анализировали сигналы GFP методом проточной цитометрии на 2-й, 5-й и 7-й день. Проявлялась различная эффективность продукции у различных систем, причем NILV был ближе всего к WT по продукции. Во всех случаях наблюдалось четкое восстановление встраивающей активности при проведении транскомплементации с WT-HIV_IN. Проверку попадания IN в систему транскомплементации проводили методом вестерн-блоттинга. WT: лентивирус, полученный с IN дикого типа; NILV: лентивирус, полученный с невстраивающейся IN, несущей две мутации в своем каталитическом центре; TAA: лентивирус, полученный с дефектной IN, у которой не экспрессируется белок вследствие наличия стоп-кодона в начале кодирующей последовательности IN; TAAx3: лентивирус, полученный с дефектной IN, у которой не экспрессируется белок вследствие наличия 3 последовательных стоп-кодонов в начале кодирующей последовательности IN; Delta-IN: лентивирус, полученный с дефектной IN, у которой была удалена кодирующая последовательность IN; Delta-IN_cPPT: лентивирус, полученный с дефектной IN, у которой кодирующая последовательность IN была заменена на последовательность центрального полипиримидинового тракта (cPPT); +VPR-IN: лентивирус с транскомплементацией IN, слитой с VPR на С-конце.
Раскрытие сущности изобретения
I Определения
В настоящем изобретении формы единственного числа включают значения и единственного, и множественного числа, если из контекста четко не следует иначе. Так, например, ссылка на “средство” включает и одно средство, и несколько таких средств.
Термины “нуклеиновая кислота”, “полинуклеотид” и “олигонуклеотид” применяются взаимозаменяемо и означают дезоксирибонуклеотидный либо рибонуклеотидный полимер в линейной или кольцевой конформации, а также в одноцепочечной или двухцепочечной форме. Для целей настоящего изобретения эти термины не должны рассматриваться как ограничивающие в отношении длины полимера. Эти термины могут охватывать известные аналоги природных нуклеотидов, а также нуклеотиды, модифицированные по группировкам оснований, сахаров и/или фосфата (например, фосфоротиоатным остовам). Обычно аналоги конкретных нуклеотидов имеют такую же специфичность спаривания оснований, т.е. аналог A будет образовывать пару с T.
Термины “полипептид”, “пептид” и “белок” применяются взаимозаменяемо для обозначения полимеров из аминокислотных остатков. Эти термины также применяются к таким аминокислотным полимерам, в которых одна или несколько аминокислот являются химическими аналогами или модифицированными производными соответствующих природных аминокислот.
Термин “связывающий(ся) белок” в настоящем изобретении означает такой белок, который способен нековалентно связываться с другой молекулой. Связывающийся белок может связываться, к примеру, с молекулой ДНК (ДНК-связывающий белок), молекулой РНК (РНК-связывающий белок) и/или с молекулой белка (белок-связывающий белок). В случае белок-связывающих белков они могут связываться сами с собой (образуя гомодимеры, гомотримеры и т.д.) и/или с одной или несколькими молекулами другого белка или белков. Связывающий белок может обладать более чем одним типом связывающей активности. Например, цинк-пальцевые белки обладают ДНК-связывающей, РНК-связывающей и белок-связывающей активностью.
Термин “цинк-пальцевый белок” в настоящем изобретении означает такой белок или домен в более крупном белке, который связывается с ДНК специфичным для последовательности образом посредством одного или нескольких цинковых пальцев, которые представляют собой участки аминокислотной последовательности в связывающем домене цинк-пальцевого белка, структура которых стабилизируется за счет координации иона цинка. Термин “цинк-пальцевый белок” часто сокращается как ZFP.
Термин “цинк-пальцевые нуклеазы” относится к искусственным рестрикционным ферментам, полученным путем слияния ДНК-связывающего домена типа цинкового пальца с ДНК-расщепляющим доменом. Домены типа цинковых пальцев могут быть сконструированы так, чтобы они садились на определенные желательные последовательности ДНК, что позволяет цинк-пальцевым нуклеазам садиться на уникальные последовательности в сложных геномах. Цинк-пальцевые нуклеазы часто сокращаются как ZFN или ZNP.
Термин “последовательность нуклеиновой кислоты” или “последовательность полинуклеотида” или “последовательность гена” в настоящем изобретении означает последовательность нуклеотидов любой длины, которая может представлять собой ДНК или РНК; может быть линейной, кольцевой или разветвленной и может быть одноцепочечной или двухцепочечной.
Термин “аминокислотная последовательность” или “полипептид” или “белок” в настоящем изобретении означает полимер из аминокислотных остатков. Если не указано иначе, полимер из аминокислотных остатков может быть любой длины.
Термин “экзогенные” в настоящем изобретении означает такие молекулы, которые обычно не присутствуют в клетках, но могут быть введены в клетки одним или несколькими генетическими, биохимическими или другими методами. Обычно присутствие в клетках определяется с учетом конкретной стадии развития и окружающей среды клеток. Так, к примеру, молекула, которая присутствует только во время эмбрионального развития мышцы, является экзогенной молекулой по отношению к взрослым мышечным клеткам. Точно так же молекула, индуцированная тепловым шоком, является экзогенной молекулой по отношению к клеткам, не подвергавшимся тепловому шоку. Экзогенные молекулы могут включать, к примеру, функционирующие версии неисправных эндогенных молекул или неисправные версии нормально функционирующих эндогенных молекул.
Напротив, “эндогенные” молекулы – это молекулы, которые обычно присутствуют в определенных клетках на определенной стадии развития при определенных условиях окружающей среды. Например, эндогенная нуклеиновая кислота может включать хромосомы, геном митохондрий, хлоропластов или других органелл либо природную эписомальную нуклеиновую кислоту. Другие эндогенные молекулы могут включать белки, к примеру, факторы транскрипции и ферменты.
“Сайт мишени” или “последовательность мишени” – это такая последовательность, которая определяет ту часть нуклеиновой кислоты или полипептида, с которой будет связываться связывающаяся молекула при наличии достаточных условий для связывания. Например, последовательность 5′-GAATTC-3′ является сайтом мишени для рестрикционной эндонуклеазы EcoRI.
Термин “слитые” в настоящем изобретении означает такие молекулы, в которых соединяются две или несколько молекул субъединиц, предпочтительно ковалентно. Молекулы субъединиц могут быть одного и того же химического типа или же разных химических типов молекул.
Термин “слитый белок” в настоящем изобретении означает гибридный белок, который содержит белковые домены по меньшей мере из двух разных белков. Один из белков может находиться в N-концевой части слитого белка либо в C-концевой части белка, образуя при этом “N-концевой слитый белок” или “C-концевой слитый белок”, соответственно.
Термины “ген” или “геном” в настоящем изобретении включают участки ДНК, кодирующие продукты генов, а также те участки ДНК, которые регулируют вырабатывание продуктов генов, независимо от того, примыкают ли такие регуляторные последовательности к кодирующим и/или транскрибируемым последовательностям. Соответственно, гены включают, необязательно без ограничения, последовательности промоторов, терминаторы, регуляторные последовательности трансляции типа сайтов связывания рибосом и сайтов для входа внутрь рибосом, энхансеры, сайленсеры, инсуляторы, граничные элементы, источники начала репликации, сайты прикрепления к матриксу и участки контроля локусов.
Термин “эукариотические” клетки включает, без ограничения, грибковые клетки (типа дрожжевых), растительные клетки, животные клетки, клетки млекопитающих и клетки человека (например, T-клетки).
Термин “соединяются” в настоящем изобретении означает такое соседство двух и более компонентов (типа элементов последовательности), при котором эти компоненты располагаются так, что оба компонента функционируют нормально и допускают возможность того, что по крайней мере один из компонентов может выполнять функцию, которая оказывает действие хотя бы на один из других компонентов.
“Функциональный фрагмент” белка, полипептида или нуклеиновой кислоты – это такой белок, полипептид или нуклеиновая кислота, соответственно, последовательность которых не идентична полноразмерному белку, полипептиду или нуклеиновой кислоте, но сохраняет ту же функцию, что и полноразмерный белок, полипептид или нуклеиновая кислота. Функциональный фрагмент может содержать больше, меньше или такое же количество остатков, что и соответствующая нативная молекула, и/или может содержать одну или несколько замен аминокислот или нуклеотидов.
Термин “трансфекция” в настоящем изобретении означает введение нуклеиновых кислот (ДНК или РНК) в эукариотические или прокариотические клетки либо организмы.
Термин “расщепление” в настоящем изобретении означает разрыв ковалентного остова молекулы ДНК. Расщепление можно инициировать различными методами, включая, без ограничения, ферментативный или химический гидролиз фосфодиэфирной связи. Возможно как одноцепочечное расщепление, так и двухцепочечное расщепление, причем двухцепочечное расщепление может происходить в результате двух отдельных событий одноцепочечного расщепления. Расщепление ДНК может приводить к образованию тупых концов либо смещенных концов. В некоторых воплощениях для направленного расщепления двухцепочечной ДНК применяются слитые полипептиды.
Термин “интеграза” в настоящем изобретении означает фермент, который вырабатывается вирусом и обеспечивает встраивание генетического материала в ДНК, например, в геномную ДНК инфицированных клеток.
Термин “специфичность” в настоящем изобретении означает способность избирательно связываться с последовательностью, которая имеет некоторую степень идентичности последовательности с выбранной последовательностью.
Термины “вставка” и “встраивание” в настоящем изобретении означают добавление одной последовательности нуклеиновой кислоты во вторую последовательность нуклеиновой кислоты или в геном.
Термины “специфическая”, “сайт-специфичная”, “целевая” и “направленная” в отношении вставки или встраивания применяются здесь взаимозаменяемо для обозначения вставки одной нуклеиновой кислоты в определенный сайт второй нуклеиновой кислоты или генома. Термины “случайная”, “ненаправленная” и “неприцельная” обозначают неспецифические и непреднамеренные генетические вставки. Термины “общее” или “полное” относятся к общему количеству вставок.
Термин “мутация” в настоящем изобретении означает замену одного остатка в последовательности, например, последовательности нуклеиновой кислоты или аминокислотной последовательности, другим остатком, либо делецию или встраивание одного или нескольких остатков в последовательность. Мутации обычно описываются здесь путем идентификации исходного остатка с указанием положения этого остатка в последовательности и идентификации вновь замещенного остатка. В данной области хорошо известны различные методы проведения аминокислотных замен (мутаций), предусмотренных здесь, которые приведены, к примеру, в Green and Sambrook. Molecular Cloning: A Laboratory Manual (4th ed., Cold Spring Harbor Laboratory Press, Cold Spring Harbor, N.Y. (2012)).
Термин “транспозаза” в настоящем изобретении означает такой фермент, который связывается с концом транспозона и катализирует его перемещение в другую часть генома по механизму вырезания и встраивания или по механизму репликативной транспозиции.
Термин “модифицированная” в настоящем изобретении означает такую последовательность белка или нуклеиновой кислоты, которая отличается от соответствующей немодифицированной последовательности белка или нуклеиновой кислоты.
Термин “линкер” в настоящем изобретении обозначает химическую группу или молекулу, соединяющую две соседние молекулы или группировки.
Термины “вектор” и “плазмида” в настоящем изобретении означают такие полинуклеотиды, которые могут нести, например, второй представляющий интерес полинуклеотид, и могут, например, переносить последовательности генов в клетки мишени. Таким образом, эти термины включают клонирующие и экспрессирующие носители, а также встраивающие векторы. В частности, термин “экспрессирующий вектор” в настоящем изобретении означает любой полинуклеотид, способный управлять экспрессией нуклеиновой кислоты. В некоторых аспектах термины “вектор” и “плазмида” применяются взаимозаменяемо с термином “конструкция из нуклеиновой кислоты”.
Термин “степень идентичности” в настоящем изобретении означает степень идентичности двух последовательностей, будь то последовательностей нуклеиновых кислот или аминокислотных последовательностей, и представляет количество точных совпадений между двумя выровненными последовательностями, деленное на длину более короткой последовательности и умноженное на 100.
Термины “рекомбинантные” или “сконструированные” в настоящем изобретении означают такие последовательности белков или нуклеиновых кислот, которые были созданы искусственно.
Термин “субъект” в настоящем изобретении означает индивидуальный организм, к примеру, индивидуальное млекопитающее. В некоторых воплощениях субъектом является человек. В некоторых воплощениях субъектами являются другие млекопитающие, а не человек. В некоторых воплощениях субъектами являются другие приматы, а не человек. В некоторых воплощениях субъектами являются грызуны. В некоторых воплощениях субъектами являются овцы, козы, крупный рогатый скот, кошки или собаки. В некоторых воплощениях субъектами являются позвоночные, амфибии, рептилии, рыбы, насекомые, мухи или нематоды. В некоторых воплощениях субъектами являются исследовательские животные.
Термины “лечение”, “лечить” и “лечащий” означают клиническое вмешательство, направленное на регрессию, ослабление, замедление возникновения или подавление развития заболевания или расстройства либо одного или нескольких его симптомов, как описано здесь. В настоящем изобретении термины “лечение”, “лечить” и “лечащий” означают клиническое вмешательство, направленное на регрессию, ослабление, замедление возникновения или подавление развития заболевания или расстройства либо одного или нескольких его симптомов, как описано здесь. В некоторых воплощениях лечение может проводиться после появления одного или нескольких симптомов и/или после диагноза заболевания. В других воплощениях лечение может проводиться и при отсутствии симптомов, например, для предотвращения, снижения вероятности возникновения или замедления появления симптомов либо для подавления возникновения или прогрессирования заболевания. Например, лечение может назначаться восприимчивым индивидам еще до появления симптомов (например, с учетом симптомов в анамнезе и/или в свете генетических или других факторов восприимчивости). Лечение может продолжаться и после прохождения симптомов, к примеру, для предотвращения или замедления их рецидива.
II Конструкции из нуклеиновой кислоты
Направленное редактирование последовательностей нуклеиновой кислоты, например, введение определенных модификаций (например, включение экзогенной нуклеиновой кислоты) в геномную ДНК, является перспективным подходом к лечению генетических заболеваний человека. С этой целью авторы изобретения стремились получить усовершенствованные конструкции из нуклеиновой кислоты для применения при редактировании генома, которые будут высокоэффективными при введении требуемых модификаций; обладать минимальной внецелевой активностью; и способными программироваться с тем, чтобы редактировать определенный сайт в геноме человека.
В некоторых аспектах настоящего изобретения предусмотрены конструкции из нуклеиновой кислоты для применения в целях улучшения сайт-специфического встраивания в геном экзогенной нуклеиновой кислоты, например, представляющего интерес гена (GOI). В некоторых воплощениях GOI является терапевтическим геном, например, геном, кодирующим терапевтический белок. Примеры представляющих интерес терапевтических генов включают ген CFTR (регулятора трансмембранной проводимости при кистозном фиброзе) для лечения муковисцидоза; ген SMN1 (ген выживаемости двигательных нейронов-1) для лечения спинальной мышечной атрофии (SMA); вариант G171V гена LRP5 (белка 5, связанного с рецептором LDL) для предотвращения остеопороза и переломов костей; и вариант A673T гена АРР (белка-предшественника бета-амилоида) для снижения предрасположенности к болезни Альцгеймера.
В некоторых воплощениях экзогенная нуклеиновая кислота для вставки (например, GOI) может иметь длину до 10 т.п.н., до 15 т.п.н., до 20 т.п.н., до 25 т.п.н., до 30 т.п.н., до 35 т.п.н. или до 40 т.п.н.
В некоторых воплощениях последовательность полинуклеотида, кодирующего ДНК-связывающий белок, обеспечивающий встраивание экзогенной нуклеиновой кислоты в геном, включает интегразу либо интегразу, модифицированную относительно интегразы дикого типа, а экзогенная нуклеиновая кислота для встраивание может иметь длину до 10 т.п.н., до 15 т.п.н. или до 20 т.п.н., например, от 1 т.п.н. до 20 т.п.н., от 1 т.п.н. до 19 т.п.н., от 1 до 18 т.п.н., от 1 т.п.н. до 17 т.п.н., от 1 т.п.н. до 16 т.п.н. или от 1 т.п.н. до 15 т.п.н.
В некоторых воплощениях последовательность полинуклеотида, кодирующего второй ДНК-связывающий белок, обеспечивающий встраивание экзогенной нуклеиновой кислоты в геном, включает транспозазу или транспозазу, модифицированную относительно транспозазы дикого типа, а экзогенная нуклеиновая кислота для вставки может иметь длину до 10 т.п.н., до 15 т.п.н., до 20 т.п.н., до 25 т.п.н., до 30 т.п.н., до 35 т.п.н. или до 40 т.п.н., например, от 1 т.п.н. до 40 т.п.н., от 1 т.п.н. до 39 т.п.н., от 1 до 38 т.п.н., от 1 т.п.н. до 37 т.п.н., от 1 т.п.н. до 36 т.п.н. или от 1 т.п.н. до 35 т.п.н.
В некоторых воплощениях конструкция из нуклеиновой кислоты включает последовательность полинуклеотида, кодирующего первый ДНК-связывающий белок, например, полипептид для редактирования генов, и последовательность полинуклеотида, кодирующего второй ДНК-связывающий белок, например, интегразу или транспозазу, причем конструкция нуклеиновой кислоты кодирует первый и второй связывающие белки в виде слитого белка. В некоторых воплощениях конструкция из нуклеиновой кислоты также включает последовательность нуклеиновой кислоты, кодирующей линкер между первым и вторым связывающим белком. В некоторых воплощениях конструкция из нуклеиновой кислоты кодирует слитый белок, обеспечивающий и/или способствующий сайт-специфической вставке экзогенной нуклеиновой кислоты в геном. В некоторых воплощениях первый или второй связывающий белок представляет собой интегразу, модифицированную относительно дикого типа. В некоторых воплощениях первый или второй связывающий белок представляет собой транспозазу, модифицированную относительно дикого типа. В некоторых воплощениях предусмотрены векторы или плазмиды, содержащие конструкции из нуклеиновой кислоты по изобретению. В некоторых воплощениях конструкция из нуклеиновой кислоты по изобретению кодирует слитый белок, который улучшает специфичность встраивания нуклеиновой кислоты, например, GOI, в геном. В некоторых воплощениях слитый белок и экзогенная нуклеиновая кислота вводятся в клетки с помощью лентивирусных частиц.
В некоторых воплощениях первый и второй связывающие белки находятся в разных конструкциях из нуклеиновой кислоты, например, транспозаза или интеграза (например, транспозаза и/или интеграза, модифицированная относительно дикого типа) находится в другой конструкции из нуклеиновой кислоты, отдельно от Cas9 или ZFP.
В некоторых аспектах предусмотрены векторы или плазмиды, содержащие приведенные здесь конструкции из нуклеиновых кислот. В некоторых воплощениях плазмиды, содержащие конструкции из нуклеиновой кислоты, являются упаковочными плазмидами. В некоторых воплощениях плазмиды, содержащие конструкции из нуклеиновой кислоты, дополнительно содержат полинуклеотид, кодирующий капсидные белки, например, gag и pol. В некоторых воплощениях (i) плазмиды, содержащие конструкции из нуклеиновой кислоты, комбинируют с (ii) плазмидами, содержащими полинуклеотид, кодирующий белки оболочки вируса (плазмидами для оболочки); и (iii) плазмидами, содержащими последовательность экзогенной нуклеиновой кислоты (например, GOI), причем при введении этой комбинации в клетки продуцирующей линии (например, эукариотические клетки, прокариотические клетки и/или клеточные линии) вырабатываются вирусные частицы, содержащие экзогенную нуклеиновую кислоту, например, GOI, и слитый белок, включающий и первый, и второй связывающий белок.
В некоторых воплощениях (i) плазмиды, содержащие конструкции из нуклеиновой кислоты, комбинируют с (ii) плазмидами, содержащими конструкции нуклеиновой кислоты, дополнительно содержащие полинуклеотид, кодирующий капсидные белки, например, gag и pol (упаковочными плазмидами, лишенными функциональной интегразы); (iii) плазмидами, содержащими полинуклеотид, кодирующий белки оболочки вируса (плазмидами для оболочки); и (iv) плазмидами, содержащими последовательность экзогенной нуклеиновой кислоты (например, GOI), причем при введении этой комбинации в клетки продуцирующей линии (например, эукариотические клетки, прокариотические клетки и/или клеточные линии) вырабатываются вирусные частицы, содержащие экзогенную нуклеиновую кислоту, например, GOI, и слитый белок, включающий и первый, и второй связывающий белок.
Конструкции из нуклеиновой кислоты содержат последовательность первого полинуклеотида, кодирующего первый ДНК-связывающий белок, сконструированный для связывания определенной последовательности ДНК, последовательность второго полинуклеотида, кодирующего второй ДНК-связывающий белок, обеспечивающий встраивание экзогенной нуклеиновой кислоты в геном, причем второй ДНК-связывающий белок представляет собой интегразу или транспозазу (например, транспозазу и/или интегразу, модифицированную относительно дикого типа), и последовательность третьего полинуклеотида, включающего последовательность нуклеиновой кислоты, кодирующей линкер между первым и вторым полинуклеотидом. В некоторых воплощениях первый ДНК-связывающий белок представляет собой цинк-пальцевый белок или белок Cas9.
В некоторых воплощениях конструкция из нуклеиновой кислоты включает линкер, выбранный из группы, состоящей из (GGS)n, (GGGGS)n (SEQ ID NO: 133), (G)n, (EAAAK)n (SEQ ID NO: 134), линкеров на основе XTEN или мотивов (XP)n либо их комбинаций, где n независимо означает целое число от 1 до 50. В некоторых воплощениях нуклеиновая кислота кодирует линкер, включающий последовательность XTEN или последовательность GGS. В некоторых воплощениях последовательность нуклеиновой кислоты линкера имеет длину от 3 до 150 нуклеотидов. В некоторых воплощениях линкер имеет длину от 12 до 24 аминокислот либо от 36 до 72 нуклеотидов. В некоторых воплощениях конструкция из нуклеиновой кислоты включает последовательность нуклеиновой кислоты линкера длиной от 6 до 120, от 6 до 90, от 6 до 78, от 6 до 72, от 9 до 120, от 9 до 90, от 9 до 78, от 9 до 72, от 12 до 120, от 12 до 90, от 12 до 78, от 12 до 72, от 15 до 120, от 15 до 90, от 15 до 78, от 15 до 72, от 18 до 120, от 18 до 90, от 18 до 78, от 18 до 72, от 21 до 120, от 21 до 90, от 21 до 78, от 21 до 72, от 24 до 120, от 24 до 90, от 24 до 78, от 24 до 72, от 27 до 120, от 27 до 90, от 27 до 78, от 27 до 72, от 30 до 120, от 30 до 90, от 30 до 78, от 30 до 72, от 33 до 120, от 33 до 90, от 33 до 78, от 33 до 72, от 36 до 120, от 36 до 90, от 36 до 78 или от 36 до 72 нуклеотидов. В некоторых воплощениях нуклеиновая кислота, кодирующая линкер, имеет длину от 9 до 150 нуклеотидов. В некоторых воплощениях цинк-пальцевый белок соединяется с модифицированной интегразой по изобретению с помощью линкера, включающего последовательность GGS. В некоторых воплощениях линкер имеет длину от 1 до 50 аминокислот. В некоторых воплощениях линкер имеет длину от 3 до 40, от 3 до 30, от 3 до 29, от 3 до 24, от 4 до 40, от 4 до 30, от 4 до 29, от 4 до 24, от 5 до 40, от 5 до 30, от 5 до 29, от 5 до 24, от 6 до 40, от 6 до 30, от 6 до 29, от 6 до 24, от 7 до 40, от 7 до 30, от 7 до 29, от 7 до 24, от 8 до 40, от 8 до 30, от 8 до 29, от 8 до 24, от 9 до 40, от 9 до 30, от 9 до 29, от 9 до 24, от 10 до 40, от 10 до 30, от 10 до 29, от 10 до 24, от 11 до 40, от 11 до 30, от 11 до 29, от 11 до 24, от 12 до 40, от 12 до 30, от 12 до 29 или от 12 до 24 аминокислот.
В некоторых воплощениях 3′-конец последовательности первого полинуклеотида соединяется с 5′-концом последовательности второго полинуклеотида нуклеиновой кислотой, кодирующей линкер. В некоторых воплощениях 5′-конец последовательности первого полинуклеотида соединяется с 3′-концом последовательности второго полинуклеотида нуклеиновой кислотой, кодирующей линкер. В некоторых воплощениях 3′-конец белка Cas9 соединяется с 5′-концом транспозазы при помощи линкера. В некоторых воплощениях 5′-конец белка Cas9 соединяется с 3′-концом транспозазы при помощи линкера. В некоторых воплощениях 3′-конец цинк-пальцевого белка соединяется с 5′-концом интегразы при помощи линкера. В некоторых воплощениях 5′-конец цинк-пальцевого белка соединяется с 3′-концом интегразы при помощи линкера.
В некоторых воплощениях линкер не нужен, так как модифицированная интеграза или модифицированная транспозаза экспрессируется из другой плазмиды, отдельно от Cas9 или ZFP.
В некоторых аспектах изобретения предусмотрены векторы или плазмиды (например, экспрессирующие векторы или упаковочные векторы, содержащие конструкции из нуклеиновой кислоты по изобретению, подходящие для экспрессии в клетках хозяина, например, в клетках млекопитающих, дрожжевых клетках, клетках насекомых, растительных клетках, грибковых клетках или клетках водорослей.
В некоторых воплощениях конструкции из нуклеиновой кислоты содержат: (a) последовательность первого полинуклеотида, включающего нуклеиновую кислоту, кодирующую первый ДНК-связывающий белок, сконструированный для связывания с определенной последовательностью геномной ДНК в геноме, причем первый ДНК-связывающий белок представляет собой цинк-пальцевый белок или белок Cas9; (b) последовательность второго полинуклеотида, включающего нуклеиновую кислоту, кодирующую второй ДНК-связывающий белок, который обеспечивает встраивание экзогенной нуклеиновой кислоты в геном, причем второй ДНК-связывающий белок представляет собой (i) гиперактивную транспозазу PiggyBac или модифицированную гиперактивную PiggyBac с улучшенной специфичностью встраивания экзогенной нуклеиновой кислоты в геном по сравнению с гиперактивной PiggyBac либо (ii) интегразу вируса иммунодефицита человека (ВИЧ) или модифицированную интегразу ВИЧ с улучшенной специфичностью встраивания экзогенной нуклеиновой кислоты в геном по сравнению с интегразой ВИЧ; и (c) необязательно последовательность полинуклеотида, включающего нуклеиновую кислоту, кодирующую линкер; при этом конструкция из нуклеиновой кислоты кодирует слитый белок, включающий первый ДНК-связывающий белок, второй ДНК-связывающий белок и необязательно линкер между первым ДНК-связывающим белком и вторым ДНК-связывающим белком; причем слитый белок обеспечивает встраивание экзогенной нуклеиновой кислоты в определенный сайт генома.
В одном воплощении (a) первый ДНК-связывающий белок представляет собой белок Cas9 или цинк-пальцевый белок; а (b) второй ДНК-связывающий белок представляет собой гиперактивную транспозазу PiggyBac или модифицированную гиперактивную транспозазу PiggyBac с улучшенной специфичностью встраивания экзогенной нуклеиновой кислоты в геном по сравнению с гиперактивной транспозазой PiggyBac.
В другом воплощении (a) первый ДНК-связывающий белок представляет собой белок Cas9 или цинк-пальцевый белок; а (b) второй ДНК-связывающий белок представляет собой интегразу ВИЧ или модифицированную интегразу ВИЧ с улучшенной специфичностью встраивания экзогенной нуклеиновой кислоты в геном по сравнению с интегразой ВИЧ.
В некоторых воплощениях белок Cas9 является таким, как описано в данном изобретении, в частности, выбранным из группы, состоящей из Cas9 человека, никазы Cas9 и мертвого Cas9, более предпочтительно это Cas9 человека или никаза Cas9.
В одном воплощении при использовании dCas9 второй ДНК-связывающий белок не является каталитическим доменом рекомбиназы Gin, Hin или Tn3 либо расщепляющим ДНК доменом FokI. Таким рекомбиназам и FoKI необходим известный сайт (акцепторная последовательность в геноме), чтобы осуществлять встраивание; поэтому возможности посадки на сайты гораздо более ограничены; к тому же для их функциональности требуется образование димеров, например, Gin.
В другом воплощении цинк-пальцевый белок является таким, как описано
в данном изобретении, предпочтительно это цинк-пальцевый белок C2H2, содержащий 6 связывающих доменов.
В следующем воплощении линкер является таким, как описано в данном изобретении, а предпочтительно линкер включает последовательность XTEN (например, SEQ ID NO: 61, кодируемую по SEQ ID NO: 60) или последовательность GGS, более предпочтительно GGSx3 (SEQ ID NO: 49, кодируется по SEQ ID NO: 48), GGSx4 (SEQ ID NO: 51, кодируется по SEQ ID NO: 50), GGSx5 (SEQ ID NO: 53, кодируется по SEQ ID NO: 52), GGSx6 (SEQ ID NO: 55, кодируется по SEQ ID NO: 54), GGSx7 (SEQ ID NO: 57, кодируется по SEQ ID NO: 56) или GGSx8 (SEQ ID NO: 59, кодируется по SEQ ID NO: 58).
В следующем воплощении 3′-конец последовательности первого полинуклеотида соединяется с 5′-концом второго полинуклеотида.
В некоторых воплощениях модифицированная гиперактивная транспозаза PiggyBac является такой, как описано в данном изобретении. В других воплощениях модифицированная интеграза ВИЧ является такой, как описано в изобретении.
В других воплощениях не используется линкер. Вместо этого, например, последовательности первого и/или второго полинуклеотида содержат нуклеиновые кислоты, кодирующие первый и второй ДНК-связывающий белок, а также дополнительные нуклеиновые кислоты по крайней мере на одном из своих концов, которые выполняют функцию линкера.
В одном воплощении (a) первый ДНК-связывающий белок представляет собой белок Cas9 или цинк-пальцевый белок, а (b) второй ДНК-связывающий белок представляет собой гиперактивную транспозазу PiggyBac или модифицированную гиперактивную PiggyBac с улучшенной специфичностью встраивания экзогенной нуклеиновой кислоты в геном по сравнению с гиперактивной PiggyBac, при этом конструкция из нуклеиновой кислоты содержит (c) последовательность полинуклеотида, включающего нуклеиновую кислоту, кодирующую линкер, включающий последовательность XTEN или последовательность GGS, причем 3′-конец последовательности первого полинуклеотида соединяется с 5′-концом второго полинуклеотида.
В одном воплощении (a) первый ДНК-связывающий белок представляет собой белок Cas9, а (b) второй ДНК-связывающий белок представляет собой гиперактивную транспозазу PiggyBac или модифицированную гиперактивную PiggyBac при условии, что, когда Cas9 является неактивным белком (dCas9), то линкером не является KLAGGAP AVGGGPK (SEQ ID NO: 130).
В одном воплощении (a) первый ДНК-связывающий белок представляет собой цинк-пальцевый белок, а (b) второй ДНК-связывающий белок представляет собой гиперактивную транспозазу PiggyBac или модифицированную гиперактивную PiggyBac, причем цинк-пальцевый белок способен распознавать множественные сайты узнавания, поскольку, как изложено в настоящем изобретении, связывающий домен цинк-пальцевого белка может быть сконструирован для связывания с выбранной последовательностью.
В одном воплощении (a) первый ДНК-связывающий белок представляет собой цинк-пальцевый белок, (b) второй ДНК-связывающий белок представляет собой гиперактивную транспозазу PiggyBac или модифицированную гиперактивную PiggyBac, а линкер представляет собой XTEN.
В одном воплощении (a) первый ДНК-связывающий белок представляет собой цинк-пальцевый белок, а (b) второй ДНК-связывающий белок представляет собой гиперактивную транспозазу PiggyBac или модифицированную гиперактивную PiggyBac, причем цинк-пальцевый белок не содержит ДНК-связывающего домена Gal4. Gal4 связывается с CGG-N11-CCG, где N может быть любым основанием. Этот белок является положительным регулятором экспрессии генов, индуцируемых галактозой, типа GAL1, GAL2, GAL7, GAL10 и MEL1, которые кодируют ферменты, используемые для превращения галактозы в глюкозу. Он распознает последовательность из 17 пар оснований (5′-CGGRNNRCYNYNCNCCG-3′) (SEQ ID NO: 135) в вышележащей активирующей последовательности (UAS-G) этих генов. Таким образом, Gal4 распознает короткую и очень частую последовательность в геноме, поэтому он не является сайт-специфичным. В предпочтительном воплощении цинк-связывающий белок содержит ДНК-связывающий домен Gal4, сконструированный так, чтобы он был сайт-специфичным.
В одном воплощении (a) первый ДНК-связывающий белок представляет собой цинк-пальцевый белок, а (b) второй ДНК-связывающий белок представляет собой гиперактивную транспозазу PiggyBac или модифицированную гиперактивную PiggyBac при условии, что линкером не является EFGGGGSGGGGSGGGGSQF (SEQ ID NO: 131).
В одном воплощении (a) первый ДНК-связывающий белок представляет собой белок Cas9 или цинк-пальцевый белок, а (b) второй ДНК-связывающий белок представляет собой интегразу ВИЧ или модифицированную интегразу ВИЧ с улучшенной специфичностью встраивания экзогенной нуклеиновой кислоты в геном по сравнению с интегразой ВИЧ, при этом конструкция из нуклеиновой кислоты содержит (c) последовательность полинуклеотида, включающего нуклеиновую кислоту, кодирующую линкер, включающий последовательность XTEN или последовательность GGS, причем 3′-конец последовательности первого полинуклеотида соединяется с 5′-концом второго полинуклеотида.
В некоторых воплощениях конструкция из нуклеиновой кислоты находится в виде ДНК или РНК.
Также предусмотрены векторы, содержащие любые конструкции из нуклеиновой кислоты, предусмотренные в изобретении. В частности, эти векторы подходят для экспрессии в клетках млекопитающих, дрожжевых клетках, клетках насекомых, клетках растений, грибковых клетках или клетках водорослей. Также предусмотрены клетки-хозяева, содержащие любые конструкции из нуклеиновой кислоты или векторы, предусмотренные в изобретении.
III Интеграза и модифицированная интеграза
Интеграза является ключевым ферментом для стабильного встраивания вирусного генома в клетки хозяина, но интеграза также связана со вставочным мутагенезом, так как место встраивания под действием интегразы дикого типа непредсказуемо. Было показано, что предпочтительно встраивание идет в сильно транскрибируемые гены, что повышает риск мутации важных генов и регуляторов. В общем, интеграза ВИЧ-1 состоит из N-концевого домена (NTD), каталитического ядра (CCD) и C-концевого домена (CTD). NTD служит для связывания и координации катиона Zn2+ в качестве важного кофактора, тогда как CTD служит для связывания ДНК. Домен CCD образует каталитическое ядро, в котором происходит катализ процесса встраивания. После проникновения в клетки хозяина и обратной транскрипции РНК вирусного генома четыре молекулы интегразы образуют тетрамер и прикрепляются к концам вирусной ДНК, которая после этого именуется интасомой. Преинтеграционный комплекс (PIC) отщепляет 3′-OH-конец ДНК, образуя 5′-OH-выступ, который потребуется позже для нуклеофильной атаки на ДНК хозяина. Во время образования этого комплекса PIC переносится в ядро. После переноса в ядро PIC образует комплекс с ДНК хозяина, именуемый комплексом переноса цепи (STC). При этом оба 3′-OH-выступа вирусной ДНК атакуют два участка каркаса ДНК хозяина с интервалом в 5 нуклеотидов. Это приводит к дупликации мишени из 5 нуклеотидов. После нуклеофильной атаки вирусная ДНК встраивается, а одноцепочечные участки ДНК подвергаются репарации под действием механизмов репарации ДНК в клетках хозяина.
Настоящим изобретением предусмотрены конструкции из нуклеиновой кислоты, содержащие полинуклеотиды, кодирующие интегразы и модифицированные интегразы для встраивания экзогенной нуклеиновой кислоты в определенное место генома. В некоторых воплощениях экзогенная нуклеиновая кислота для встраивания может иметь длину до 10 т.п.н., до 15 т.п.н. или до 20 т.п.н., например, от 1 т.п.н. до 20 т.п.н., от 1 т.п.н. до 19 т.п.н., от 1 т.п.н. до 18 т.п.н., от 1 т.п.н. до 17 т.п.н., от 1 т.п.н. до 16 т.п.н. или от 1 т.п.н. до 15 т.п.н. В некоторых воплощениях последовательность полинуклеотида, кодирующего ДНК-связывающий белок, который обеспечивает встраивание экзогенной нуклеиновой кислоты в геном, включает интегразу, которая может быть модифицированной относительно интегразы дикого типа, а экзогенная нуклеиновая кислота для встраивания может иметь длину до 10 т.п.н. или до 15 т.п.н.
В некоторых аспектах изобретения предусмотрены слитые белки интегразы, разработанные с помощью описанных здесь способов и стратегий. В некоторых воплощениях изобретения предусмотрены нуклеиновые кислоты, кодирующие интегразы или модифицированные интегразы и/или содержащие их слитые белки. В некоторых воплощениях изобретения предусмотрены плазмиды или экспрессирующие векторы, содержащие такие конструкции из нуклеиновой кислоты, кодирующие интегразы или модифицированные интегразы и/или содержащие их слитые белки.
Интегразой или модифицированной интегразой по изобретению может быть любая интеграза, которая может вставить экзогенную нуклеиновую кислоту в определенное место генома. Неограничительные примеры интеграз включают интегразу ВИЧ, лентивирусную интегразу, аденовирусную интегразу, ретровирусную интегразу и интегразу вируса опухолей молочной железы мыши. В некоторых воплощениях интеграза (например, модифицированная интеграза, содержащая одну или несколько модификаций относительно дикого типа) представляет собой интегразу ВИЧ, предпочтительно интегразу ВИЧ с последовательностью, соответствующей NC_001802.1 (SEQ ID NO: 1 и 2, аминокислотная и нуклеотидная последовательность, соответственно). В некоторых воплощениях модифицированная интеграза содержит одну или несколько модификаций относительно интегразы ВИЧ дикого типа (SEQ ID NO: 1 и 2).
В некоторых воплощениях интеграза представляет собой модифицированную интегразу ВИЧ. Модифицированная интеграза ВИЧ может содержать мутации одной или нескольких аминокислот, выбранных из числа аминокислот 10, 13, 64, 94, 116, 117, 119, 120, 122, 124, 128, 152, 168, 170, 185, 231, 264, 266 или 273 в соответствии с нумерацией аминокислот по SEQ ID NO: 1. Мутации модифицированной интегразы ВИЧ могут включать одну или несколько модификаций аминокислот, приведенных в таблице 8. Мутации модифицированной интегразы ВИЧ могут включать одну или несколько модификаций аминокислот из числа D10K, E13K, D64A, D64E, G94D, G94E, G94R, G94K, D116A, D116E, N117D, N117E, N117R, N117K, S119A, S119P, S119T, S119G, S119D, S119E, S119R, S119K, N120D, N120E, N120R, N120K, T122K, T122I, T122V, T122A, T122R, A124D, A124E, A124R, A124K, A128T, E152A, E152D, Q168L, Q168A, E170G, F185K, R231G, R231K, R231D, R231E, R231S, K264R, K266R или K273R в соответствии с нумерацией аминокислот по SEQ ID NO: 1 или SEQ ID NO: 3.
В некоторых воплощениях модифицированная интеграза может содержать одну или несколько мутаций относительно дикого типа, нарушающих связывание ДНК, например, по аминокислотам 94, 117, 119, 120, 124 и/или 231 (например, G94D, G94E, G94R, G94K, N117D, N117E, N117R, N117K, S119A, S119P, S119T, S119G, S119D, S119E, S119R, S119K, N120D, N120E, N120R, N120K, A124D, A124E, A124R, A124K, R231G, R231K, R231D, R231E и/или R231K) в соответствии с нумерацией аминокислот по SEQ ID NO: 1 или SEQ ID NO: 4.
В некоторых воплощениях модифицированная интеграза может содержать одну или несколько мутаций относительно дикого типа, усиливающих связывание ДНК, например, по аминокислотам 94, 117, 119, 120, 122, 124 и/или 231 (например, G94D, G94E, G94R, G94K, N117D, N117E, N117R, N117K, S119A, S119P, S119T, S119G, S119D, S119E, S119R, S119K, N120D, N120E, N120R, N120K, T122K, T122I, T122V, T122A, T122R, A124D, A124E, A124R, A124K, R231G, R231K, R231D, R231E и/или R231S) в соответствии с нумерацией аминокислот по SEQ ID NO: 1 или SEQ ID NO: 5.
В некоторых воплощениях модифицированная интеграза может содержать одну или несколько мутаций относительно дикого типа по аминокислотам, участвующим в ацетилировании интегразы под действием p300, например, по аминокислотам 264, 266 и/или 273 (например, K264R, K266R и/или K273R) в соответствии с нумерацией аминокислот по SEQ ID NO: 1 или SEQ ID NO: 6.
В некоторых воплощениях модифицированная интеграза может содержать одну или несколько мутаций по сильно консервативным аминокислотам, важным для интегративной рекомбинации ретровирусов, например, по аминокислотам 10, 13, 64, 116, 128, 152, 168 и/или 170 (например, D10K, E13K, D64A, D64E, D116A, D116E, A128T, E152A, E152D, Q168L, Q168A и/или E170G) в соответствии с нумерацией аминокислот по SEQ ID NO: 1 или SEQ ID NO: 7.
В некоторых воплощениях модифицированная интеграза может содержать одну или несколько мутаций, нарушающих взаимодействие с LEDGF/p75 и нарушающих привязку хромосом и репликацию ВИЧ-1, например, по аминокислоте 168 (например, Q168L или Q168A) в соответствии с нумерацией аминокислот по SEQ ID NO: 1 или SEQ ID NO: 8.
В некоторых воплощениях модифицированная интеграза ВИЧ имеет аминокислотную последовательность, которая по меньшей мере на 80%, на 85%, на 90%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности, приведенной в SEQ ID NO: 1. В некоторых воплощениях модифицированная интеграза ВИЧ имеет аминокислотную последовательность, содержащую одну или несколько из описанных здесь модификаций относительно SEQ ID NO: 1, 3, 4, 5, 6, 7 или 8, соответственно, и остается по меньшей мере на 80%, на 85%, на 90%, на 95%, на 96%, на 97%, на 98% или на 99% идентичной последовательности, приведенной в SEQ ID NO: 1, 3, 4, 5, 6, 7 или 8, соответственно. В некоторых воплощениях модифицированная интеграза ВИЧ подвергалась отбору на высокую специфичность встраивания ДНК в геном по сравнению с интегразой ВИЧ дикого типа.
В некоторых аспектах изобретения предусмотрены векторы или плазмиды (например, экспрессирующие векторы или упаковочные векторы), содержащие конструкции из нуклеиновой кислоты, включающие интегразу или модифицированную интегразу по изобретению, подходящую для экспрессии в клетках хозяина, например, в клетках млекопитающих, дрожжевых клетках, клетках насекомых, клетках растений, грибковых клетках или клетках водорослей. В некоторых воплощениях интеграза или модифицированная интеграза экспрессируется в виде слитого белка с белком Cas9 или цинк-пальцевым белком. В некоторых воплощениях интеграза или модифицированная интеграза экспрессируется вместе с белком Cas9 или цинк-пальцевым белком из разных векторов, но вводится в одни и те же клетки. В некоторых воплощениях интеграза или модифицированная интеграза либо содержащий её слитый белок упаковывается в лентивирусные частицы для доставки в клетки.
IV Транспозаза и модифицированная транспозаза
Транспозоны – это сегменты хромосом, которые могут подвергаться транспозиции, например, это ДНК, которая может транслоцироваться как одно целое в отсутствие комплементарной последовательности в ДНК хозяина. Транспозоны могут применяться для различного рода инженерии ДНК в клетках человека. Распространенные системы транспозонов, используемые в клетках млекопитающих, включают Sleeping Beauty (SB), который был реконструирован из неактивных транспозонов, и PiggyBac (PB), выделенный из моли Trichoplusia. PiggyBac обладает большей активностью транспозиции, чем SB, и он вырезается без следа.
Транспозоны нативной ДНК обычно содержат единственный ген, кодирующий белок транспозазы, который фланкирован концевыми инвертированными повторами (ITR), несущими сайты связывания транспозазы. Во время транспозиции белок транспозазы распознает эти ITR и катализирует вырезание и последующее встраивание этого элемента в другом месте случайным образом. Более того, некоторые из этих транспозонов можно адаптировать для применения в методиках генной терапии, используя их в качестве двухкомпонентных систем, в которых плазмида содержит экспрессионную кассету, где последовательность ДНК, помещенная между ITR транспозона, может быть введена в геном хозяина под управлением котрансфицируемой плазмиды, содержащей последовательность, кодирующую фермент транспозазу либо её мРНК, синтезированную in vitro. В некоторых аспектах изобретения транспозоны применяются для эффективного обеспечения стабильного встраивания и стойкой экспрессии трансгенов типа терапевтических генов.
Настоящим изобретением предусмотрены конструкции из нуклеиновой кислоты, содержащие полинуклеотиды, кодирующие транспозазы и модифицированные транспозазы для встраивания экзогенной нуклеиновой кислоты в определенное место генома. В некоторых воплощениях экзогенная нуклеиновая кислота для вставки может иметь длину до 20 т.п.н., до 25 т.п.н., до 30 т.п.н. или до 40 т.п.н., например, от 1 т.п.н. до 40 т.п.н., от 1 т.п.н. до 39 т.п.н., от 1 до 38 т.п.н., от 1 т.п.н. до 37 т.п.н., от 1 т.п.н. до 36 т.п.н., от 1 т.п.н. до 35 т.п.н., от 1 т.п.н. до 30 т.п.н. либо от 1 т.п.н. до 25 т.п.н. В некоторых воплощениях последовательность полинуклеотида, кодирующего ДНК-связывающий белок, который обеспечивает встраивание экзогенной нуклеиновой кислоты в геном, включает транспозазу или модифицированную относительно дикого типа транспозазу, а экзогенная нуклеиновая кислота для вставки может иметь длину до 35 т.п.н. или до 40 т.п.н.
Транспозазой или модифицированной транспозазой по изобретению может быть любая транспозаза, которая может вставить экзогенную нуклеиновую кислоту в определенное место генома. В некоторых аспектах изобретения предусмотрены слитые белки транспозазы, разработанные с помощью описанных здесь способов и стратегий. В некоторых воплощениях изобретения предусмотрены нуклеиновые кислоты, кодирующие такие транспозазы или модифицированные транспозазы и/или содержащие их слитые белки. В некоторых воплощениях изобретения предусмотрены плазмиды или экспрессирующие векторы, содержащие такие конструкции из нуклеиновой кислоты, кодирующие транспозазы или модифицированные транспозазы и/или содержащие их слитые белки.
Неограничительные примеры транспозаз включают транспозазу Frog Prince, Sleeping Beauty, гиперактивную Sleeping Beauty, PiggyBac и гиперактивную PiggyBac. В некоторых воплощениях транспозаза представляет собой гиперактивную транспозазу PiggyBac по SEQ ID NO: 9 или 67 (которая в описании также именуется как hyPB или просто как PB). В некоторых воплощениях модифицированная транспозаза содержит одну или несколько модификаций относительно гиперактивной транспозазы PiggyBac (SEQ ID NO: 9).
В некоторых воплощениях транспозаза представляет собой модифицированную гиперактивную транспозазу PiggyBac. Модифицированная гиперактивная транспозаза PiggyBac может содержать мутации одной или нескольких аминокислот из числа аминокислот 245, 268, 275, 277, 287, 290, 315, 325, 341, 346, 347, 350, 351, 356, 357, 372, 375, 388, 409, 412, 432, 447, 450, 460, 461, 465, 517, 560, 564, 571, 573, 576, 586, 587, 589, 592 и 594 в соответствии с нумерацией аминокислот по SEQ ID NO: 9. Мутации модифицированной гиперактивной PiggyBac могут включать одну или несколько модификаций аминокислот, приведенных в таблице 3. Мутации модифицированной гиперактивной транспозазы PiggyBac могут включать одну или несколько модификаций аминокислот из числа R245A, D268N, R275A/R277A, K287A, K290A, K287A/K290A, R315A, G325A, R341A, D346N, N347A, N347S, T350A, S351E, S351P, S351A, K356E, N357A, R372A, K375A, R372A/K375A, R388A, K409A, K412A, K409A/K412A, K432A, D447A, D447N, D450N, R460A, K461A, R460A/K461A, W465A, S517A, T560A, S564P, S571N, S573A, K576A, H586A, I587A, M589V, S592G или F594L в соответствии с нумерацией аминокислот по SEQ ID NO: 9 или SEQ ID NO: 10.
В некоторых воплощениях модифицированная транспозаза может содержать одну или несколько мутаций относительно hyPB по аминокислотам, участвующим в консервативной каталитической триаде, например, 268 и/или 346 (например, D268N и/или D346N) в соответствии с нумерацией аминокислот по SEQ ID NO: 9 или SEQ ID NO: 11.
В некоторых воплощениях модифицированная транспозаза может содержать одну или несколько мутаций относительно hyPB по аминокислотам, важным для вырезания, например, 287, 287/290 и/или 460/461 (например, K287A, K287A/K290A и/или R460A/K461A) в соответствии с нумерацией аминокислот по SEQ ID NO: 9 или SEQ ID NO: 12.
В некоторых воплощениях модифицированная транспозаза может содержать одну или несколько мутаций относительно hyPB по аминокислотам, участвующим в посадке на мишень, например, 351, 356 и/или 379 (например, S351E, S351P, S351A и/или K356E) в соответствии с нумерацией аминокислот по SEQ ID NO: 9 или SEQ ID NO: 13.
В некоторых воплощениях модифицированная транспозаза может содержать одну или несколько мутаций относительно hyPB по аминокислотам, важным для встраивания, например, 560, 564, 571, 573, 589, 592 и/или 594 (например, T560A, S564P, S571N, S573A, M589V, S592G и/или F594L) в соответствии с нумерацией аминокислот по SEQ ID NO: 9 или SEQ ID NO: 14.
В некоторых воплощениях модифицированная транспозаза может содержать одну или несколько мутаций относительно hyPB по аминокислотам, участвующим в выравнивании, например, 325, 347, 350, 357 и/или 465 (например, G325A, N347A, N347S, T350A и/или W465A) в соответствии с нумерацией аминокислот по SEQ ID NO: 9 или SEQ ID NO: 15.
В некоторых воплощениях модифицированная транспозаза может содержать одну или несколько мутаций относительно hyPB по аминокислотам, которые весьма консервативны, например, 576 и/или 587 (например, K576A и/или I587A) в соответствии с нумерацией аминокислот по SEQ ID NO: 9 или SEQ ID NO: 16.
В некоторых воплощениях модифицированная транспозаза может содержать одну или несколько мутаций относительно hyPB по аминокислотам, участвующим в связывании Zn2+, например, 586 (например, H586A) в соответствии с нумерацией аминокислот по SEQ ID NO: 9 или SEQ ID NO: 17.
В некоторых воплощениях программируемая транспозаза может содержать одну или несколько мутаций относительно hyPB по аминокислотам, участвующим во встраивании, например, 315, 341, 372 и/или 375 (например, R315A, R341A, R372A и/или K375A) в соответствии с нумерацией аминокислот по SEQ ID NO: 9 или SEQ ID NO: 18.
В некоторых воплощениях модифицированная гиперактивная PiggyBac имеет аминокислотную последовательность, которая по меньшей мере на 85%, на 90%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности, приведенной в SEQ ID NO: 9. В некоторых воплощениях модифицированная гиперактивная PiggyBac подвергается отбору на высокую специфичность встраивания ДНК в геном по сравнению с гиперактивной PiggyBac. В некоторых воплощениях модифицированная гиперактивная PiggyBac имеет аминокислотную последовательность, содержащую одну или несколько из описанных здесь модификаций относительно SEQ ID NO: 9, 10, 11, 12, 13, 14, 15, 16, 17 или 18 и остается по меньшей мере на 80%, на 85%, на 90%, на 95%, на 96%, на 97%, на 98% или на 99% идентичной последовательности, приведенной в SEQ ID NO: 9, 10, 11, 12, 13, 14, 15, 16, 17 или 18, соответственно.
В некоторых воплощениях гиперактивная транспозаза PiggyBac кодируется нуклеиновой кислотой, последовательность которой по меньшей мере на 85%, 90%, 95%, 96%, 97%, 98%, 99% или 100% идентична последовательности SEQ ID. NO: 67. В некоторых воплощениях транспозаза SB100 кодируется нуклеиновой кислотой, последовательность которой по меньшей мере на 85%, 90%, 95%, 96%, 97%, 98%, 99% или 100% идентична последовательности SEQ ID. NO: 68.
В некоторых воплощениях транспозаза PB имеет аминокислотную последовательность, которая по меньшей мере на 85%, 90%, 95%, 96%, 97%, 98%, 99% или на 100% идентична последовательности SEQ ID NO: 72. В некоторых воплощениях транспозаза SB100 имеет аминокислотную последовательность, которая по меньшей мере на 85%, 90%, 95%, 96%, 97%, 98%, 99% или на 100% идентична последовательности SEQ ID NO: 73.
В некоторых воплощениях модифицированная транспозаза представляет собой модифицированную транспозазу Sleeping Beauty, содержащую одну или несколько мутаций. В некоторых воплощениях одна или несколько мутаций у гиперактивной транспозазы Sleeping Beauty или SB100 соответствует L25F, R36A, I42K, G59D, I212K, N245S, K252A и Q271L по SEQ ID NO: 9 или SEQ ID NO: 73.
В некоторых воплощениях модифицированная транспозаза не является мутантом Himar1C9.
В некоторых аспектах изобретения предусмотрены векторы или плазмиды (например, экспрессирующие векторы или упаковочные векторы), содержащие конструкции из нуклеиновой кислоты, включающие транспозазу или модифицированную транспозазу по изобретению, подходящую для экспрессии в клетках хозяина, например, в клетках млекопитающих, дрожжевых клетках, клетках насекомых, клетках растений, грибковых клетках или клетках водорослей. В некоторых воплощениях транспозаза или модифицированная транспозаза экспрессируется в виде слитого белка с Cas9. В некоторых воплощениях транспозаза или модифицированная транспозаза экспрессируется вместе с белком Cas9 из разных векторов, но вводится в одни и те же клетки. В некоторых воплощениях транспозаза или модифицированная транспозаза либо содержащий её слитый белок упаковывается в лентивирусные частицы для доставки в клетки.
Как показано в Примере 20, недавно разработанная библиотека мутаций гиперактивной транспозазы PiggyBac может использоваться для идентификации такой модифицированной гиперактивной PiggyBac, которая осуществляет специфические направленные транспозиции. С помощью такой библиотеки была идентифицирована модифицированная гиперактивная PiggyBac, положительная по направленной транспозиции.
В некоторых воплощениях модифицированная гиперактивная транспозаза PiggyBac может содержать мутации одной или нескольких аминокислот из числа аминокислот 245, 275, 277, 325, 347, 351, 372, 375, 388, 450, 465, 560, 564, 573, 589, 592, 594 в соответствии с нумерацией аминокислот по SEQ ID NO: 9.
В некоторых воплощениях мутации модифицированной гиперактивной PiggyBac могут включать одну или несколько модификаций аминокислот, приведенных в таблице 11.
В некоторых воплощениях мутации модифицированной гиперактивной транспозазы PiggyBac могут включать одну или несколько модификаций аминокислот из числа R245A, R275A, R277A, R275A/R277A, G325A, N347A, N347S, S351E, S351P, S351A, R372A, K375A, R388A, D450N, W465A, T560A, S564P, S573A, M589V, S592G или F594L в соответствии с нумерацией аминокислот по SEQ ID NO: 9 или SEQ ID NO: 119.
В одном воплощении модифицированная гиперактивная транспозаза PiggyBac содержит модификацию аминокислоты D450 в соответствии с нумерацией аминокислот по SEQ ID NO: 9 или SEQ ID NO: 119.
В одном воплощении модифицированная гиперактивная транспозаза PiggyBac содержит модификации аминокислот R372A, K375A и D450 в соответствии с нумерацией аминокислот по SEQ ID NO: 9 или SEQ ID NO: 119.
В одном воплощении модифицированная гиперактивная транспозаза PiggyBac содержит модификации аминокислот R245A и D450 в соответствии с нумерацией аминокислот по SEQ ID NO: 9 или SEQ ID NO: 119.
В одном воплощении модифицированная гиперактивная транспозаза PiggyBac содержит модификации аминокислот R245A, G325A и D450 в соответствии с нумерацией аминокислот по SEQ ID NO: 9 или SEQ ID NO: 119.
В одном воплощении модифицированная гиперактивная транспозаза PiggyBac содержит модификации аминокислот R245A, G325A, D450 и S573P в соответствии с нумерацией аминокислот по SEQ ID NO: 9 или SEQ ID NO: 119.
Как уже было сказано, здесь предусмотрены модифицированные гиперактивные транспозазы PiggyBac, которые могут быть слиты с приведенными здесь элементами, но также могут использоваться и сами по себе или в комбинации с другими элементами. Данные транспозазы были созданы авторами изобретения. Итак, предусмотрены модифицированные гиперактивные транспозазы PiggyBac, которые включают аминокислотную последовательность SEQ ID NO: 9, где:
i. в положении 245 находится аминокислота A,
ii. в положении 275 находится аминокислота R или A,
iii. в положении 277 находится аминокислота R или A,
iv. в положении 325 находится аминокислота A или G,
v. в положении 347 находится аминокислота N или A,
vi. в положении 351 находится аминокислота E, P или A,
vii. в положении 372 находится аминокислота R,
viii. в положении 375 находится аминокислота A,
ix. в положении 450 находится аминокислота D или N,
x. в положении 465 находится аминокислота W или A,
xi. в положении 560 находится аминокислота T или A,
xii. в положении 564 находится аминокислота P или S,
xiii. в положении 573 находится аминокислота S или A,
xiv. в положении 592 находится аминокислота G или S и
xv. в положении 594 находится аминокислота L или F.
В некоторых воплощениях модифицированная гиперактивная PiggyBac имеет аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 120, 121, 122, 123, 124, 125, 126, 127, 128 и 129.
В некоторых воплощениях модифицированная гиперактивная PiggyBac имеет аминокислотную последовательность, содержащую одну или несколько из описанных здесь модификаций относительно SEQ ID NO: 119, 120, 121, 122, 123, 124, 125, 126, 127, 128 или 129 и остается по меньшей мере на 80%, на 85%, на 90%, на 95%, на 96%, на 97%, на 98% или на 99% идентичной последовательности, приведенной в SEQ ID NO: 119, 120, 121, 122, 123, 124, 125, 126, 127, 128 или 129, соответственно. В некоторых воплощениях модифицированная гиперактивная PiggyBac подвергается отбору на высокую специфичность встраивания ДНК в геном по сравнению с гиперактивной PiggyBac.
Настоящим изобретением также предусмотрены приведенные здесь модифицированные гиперактивные транспозазы PiggyBac для применения в качестве лекарственных средств, в частности, в генной терапии ex vivo или in vivo.
V Редактирование генов с помощью Cas9 и “цинковых пальцев”
Современные инструменты инженерии генома, включая сконструированные цинк-пальцевые белки (ZFP), эффекторные нуклеазы типа активаторов транскрипции (TALEN), а в последнее время и РНК-управляемая ДНК-эндонуклеаза Cas9, осуществляют специфичное для последовательности расщепление ДНК в геноме. Такое программируемое расщепление может вызывать мутации ДНК по месту расщепления посредством негомологического соединения концов (NHEJ) или замену ДНК, окружающей сайт расщепления, посредством репарации по гомологии (HDR).
В некоторых аспектах изобретения предусмотрены конструкции из нуклеиновой кислоты, содержащие полинуклеотиды, кодирующие ДНК-связывающие белки, например, Cas9 и ZFP, сконструированные для связывания с определенной последовательностью геномной ДНК. В некоторых воплощениях такие ДНК-связывающие белки сливаются с описанной здесь модифицированной интегразой или модифицированной транспозазой для редактирования генов.
Cas9
Система CRISPR-Cas9 является высокоэффективным инструментом для инактивации или модификации генов посредством специфичных для последовательности двухцепочечных разрывов (DSB). Эти DSB распознаются клеточными механизмами ответа на повреждение ДНК и подвергаются репарации эндогенными путями репарации DSB. Преобладающим путем репарации является негомологическое соединение концов (NHEJ), которое часто приводит к небольшим вставкам и/или делециям, которые могут создавать мутации сдвига рамки считывания и нарушать функцию генов. Этот путь можно использовать для получения генетических нокаут-мутаций. С другой стороны, при наличии матрицы для репарации повреждения могут устраняться без следа посредством репарации по гомологии (HDR). Однако, несмотря на заметный прогресс, опосредованное HDR редактирование генома для внесения точных генетических модификаций намного менее эффективно, чем опосредованное NHEJ разрушение генов. Кроме того, большие замены в несколько т.п.н. посредством HDR остаются проблемными и требуют отбора и/или сортировки больших популяций клеток. Поэтому основное применение механизма HDR – локальная замена ключевых участков в генах.
Термины “Cas9” и “нуклеаза Cas9” обозначают РНК-управляемую нуклеазу, содержащую белок Cas9 либо его фрагмент (например, белок, содержащий активный или неактивный ДНК-расщепляющий домен Cas9 и/или домен, связывающий направляющую РНК, Cas9). Нуклеаза Cas9 также иногда именуется нуклеазой casn1 либо нуклеазой, ассоциированной с CRISPR (кластерами коротких палиндромным повторов с регулярными промежутками). CRISPR – это адаптивная иммунная система, обеспечивающая защиту от подвижных генетических элементов (вирусов, перемещающихся элементов и конъюгативных плазмид). Кластеры CRISPR содержат спейсеры, последовательности, комплементарные предшествующим подвижным элементам и проникающим в клетки нуклеиновым кислотам. Кластеры CRISPR подвергаются транскрипции и преобразуются в CRISPR-РНК (crRNA). В системах CRISPR типа II для правильного процессинга пре-crRNA требуется кодируемая in trans короткая РНК (tracrРНК), эндогенная рибонуклеаза 3 (rnc) и белок Cas9. TracrРНК служит проводником для процессинга пре-crRNA с помощью рибонуклеазы 3. Впоследствии Cas9/crRNA/tracrRNA эндонуклеолитически расщепляет линейную или кольцевую мишень дцДНК, комплементарную спейсеру. Нить мишени, не комплементарная crРНК, сначала разрезается эндонуклеолитически, а затем 3′-5′-экзонуклеолитически. В природе для связывания и расщепления ДНК обычно требуется белок и обе РНК. Однако можно сконструировать единую направляющую РНК (“sgRNA” или просто “направляющую РНК”) с тем, чтобы она включала аспекты как crРНК, так и tracrРНК в единый вид РНК.
Cas9 распознает короткий мотив в последовательностях повторов CRISPR (PAM, то есть прилегающий к протоспейсеру мотив), который помогает отличить «свое» от «чужого». Последовательности и структуры нуклеазы Cas9 хорошо известны специалистам в данной области. Были описаны ортологи Cas9 у различных видов, включая, без ограничения, S. pyogenes и S. thermophilus. Специалистам должны быть известны и другие подходящие нуклеазы и последовательности Cas9, исходя из данного описания, причем такие нуклеазы и последовательности Cas9 включают последовательности Cas9 из организмов и локусов, описанных в Chylinski et al., “The tracrRNA and Cas9 families of type II CRISPR-Cas immunity systems” (2013) RNA Biology 10:5, 726-737; всё содержание которой включено сюда путем ссылки.
В некоторых воплощениях нуклеаза Cas9 содержит неактивный (например, инактивированный) ДНК-расщепляющий домен. Белок Cas9 с инактивированной нуклеазой может взаимозаменяемо именоваться белком “dCas9” (от Cas9 с “мертвой” (dead) нуклеазой)). Методы получения белка Cas9 (или его фрагментов) с неактивным ДНК-расщепляющим доменом известны (например, см. Jinek et al., Science 337:816-821 (2012); Qi et al., “Repurposing CRISPR as an RNA-Guided Platform for Sequence-Specific Control of Gene Expression” (2013) Cell 28, 152(5):1173-83; всё содержание которой включено сюда путем ссылки.
Например, известно, что ДНК-расщепляющий домен Cas9 включает два субдомена: нуклеазный субдомен HNH и субдомен RuvC1. Субдомен HNH расщепляет нить, комплементарную направляющей РНК, а субдомен RuvC1 расщепляет некомплементарную нить. Мутации в этих субдоменах могут заглушать нуклеазную активность Cas9. Например, мутации D10A и H841A полностью инактивируют нуклеазную активность Cas9 S. pyogenes. Никаза Cas9 является вариантом нуклеазы Cas9, отличающимся точечной мутацией (D10A) в нуклеазном домене RuvC, что позволяет ей надрезать, но не расщеплять ДНК.
Термин “Cas9” также включает его варианты и функциональные фрагменты. В некоторых воплощениях предусмотрены белки, содержащие фрагменты Cas9. Например, в некоторых воплощениях белок содержит один из двух доменов Cas9: (1) домен Cas9 связывающий направляющую РНК; или (2) ДНК-расщепляющий домен Cas9. В некоторых воплощениях белок, содержащий Cas9 или его фрагменты, именуется “вариантом Cas9”. Вариант Cas9 имеет гомологию с Cas9 или его фрагментом. Например, вариант Cas9 может быть по меньшей мере на 70%, на 80%, на 90%, на 95%, на 96%, на 97%, на 98%, на 99%, на 99,5% или на 99,9% идентичен Cas9 дикого типа. В некоторых воплощениях вариант Cas9 содержит фрагмент Cas9 (например, домен связывающий направляющую РНК чили ДНК-расщепляющий домен), причем этот фрагмент по меньшей мере на 70%, на 80%, на 90%, на 95%, на 96%, на 97%, на 98%, на 99%, на 99,5% или на 99,9% идентичен соответствующему фрагменту Cas9 дикого типа. В некоторых воплощениях Cas9 означает Cas9 из Corynebacterium ulcerans (№ по NCBI: NC_015683.1, NC_017317.1) (SEQ ID NO: 19); Corynebacterium diphtheria (№ по NCBI: NC_016782.1, NC_016786.1) (SEQ ID NO: 20); Spiroplasma syrphidicola (№ по NCBI: NC_021284.1) (SEQ ID NO: 21); Prevotella intermedia (№ по NCBI: NC_017861.1) (SEQ ID NO: 22); Spiroplasma taiwanense (№ по NCBI: NC_021846.1) (SEQ ID NO: 23); Streptococcus iniae (№ по NCBI: NC_021314.1) (SEQ ID NO: 24); Belliella baltica (№ по NCBI: NC_018010.1) (SEQ ID NO: 25); Psychroflexus torquisi (№ по NCBI: NC_018721.1) (SEQ ID NO: 26); Streptococcus thermophilus (№ по NCBI: YP_820832.1) (SEQ ID NO: 27); Listeria innocua (№ по NCBI: NP_472073.1) (SEQ ID NO: 28); Campylobacter jejuni (№ по NCBI: YP_002344900.1) (SEQ ID NO: 29); или Neisseria meningitidis (№ по NCBI: YP_002342100.1) (SEQ ID NO: 30). В некоторых воплощениях Cas9 дикого типа соответствует Cas9 из Streptococcus pyogenes (№ по NCBI: NC_017053.1) (SEQ ID NO: 31).
Из известных белков Cas9 именно Cas9 S. pyogenes широко применяется в качестве инструмента для инженерии генома. Этот белок Cas9 представляет собой большой мультидоменный белок, содержащий два разных нуклеазных домена. В Cas9 можно вводить точечные мутации для устранения нуклеазной активности, получая “мертвый” Cas9 (dCas9), еще сохраняющий способность к связыванию ДНК запрограммированным sgRNA способом. В принципе, при слиянии с другим белком или доменом dCas9 может направлять этот белок практически на любую последовательность ДНК просто при совместной экспрессии с соответствующей sgRNA.
Настоящим изобретением предусмотрены конструкции из нуклеиновой кислоты, содержащие полинуклеотиды, кодирующие белки Cas9 для встраивания экзогенной нуклеиновой кислоты в определенный сайт генома. В некоторых аспектах изобретения предусмотрены слитые белки, включающие белок Cas9 и модифицированную интегразу либо модифицированную транспозазу по изобретению. В некоторых воплощениях изобретения предусмотрены нуклеиновые кислоты, кодирующие такие белки или слитые белки Cas9. В некоторых воплощениях предусмотрены плазмиды или экспрессирующие векторы, содержащие такие нуклеиновые кислоты.
Cas9, кодируемые приведенными здесь конструкциями из нуклеиновой кислоты, могут быть любые Cas9, которые могут связываться с определенной последовательностью геномной ДНК в геноме. Неограничительные примеры белков Cas9 включают Cas9 человека (hCas9), никазу Cas9 (nCas9), “мертвый” Cas9 (dCas9), Cas9 из Streptococcus pyogenes, Cas9 из Staphylococcus aureus, Cas12a, Cas12b, мертвый Cas9 (dCas9), их варианты и функциональные фрагменты. В некоторых воплощениях Cas9 представляет собой Cas9 человека либо его вариант или функциональный фрагмент.
В некоторых воплощениях hCas9 кодируется последовательностью нуклеиновой кислоты, которая по меньшей мере на 70%, на 75%, на 80%, на 85%, на 90%, на 95%, на 96%, на 97%, на 98%, на 99% или на 100% идентична последовательности SEQ ID NO: 64. В некоторых воплощениях nCas9 кодируется последовательностью нуклеиновой кислоты, которая по меньшей мере на 70%, на 75%, на 80%, на 85%, на 90%, на 95%, на 96%, на 97%, на 98%, на 99% или на 100% идентична последовательности SEQ ID NO: 65. В некоторых воплощениях dCas9 кодируется последовательностью нуклеиновой кислоты, которая по меньшей мере на 70%, на 75%, на 80%, на 85%, на 90%, на 95%, на 96%, на 97%, на 98%, на 99% или на 100% идентична последовательности SEQ ID NO: 66.
В некоторых воплощениях hCas9 имеет аминокислотную последовательность, которая по меньшей мере на 70%, на 75%, на 80%, на 85%, на 90%, на 95%, на 96%, на 97%, на 98%, на 99% или на 100% идентична последовательности SEQ ID NO: 69. В некоторых воплощениях nCas9 имеет аминокислотную последовательность, которая по меньшей мере на 70%, на 75%, на 80%, на 85%, на 90%, на 95%, на 96%, на 97%, на 98%, на 99% или на 100% идентична последовательности SEQ ID NO: 70. В некоторых воплощениях dCas9 имеет аминокислотную последовательность, которая по меньшей мере на 70%, на 75%, на 80%, на 85%, на 90%, на 95%, на 96%, на 97%, на 98%, на 99% или на 100% идентична последовательности SEQ ID NO: 71.
В некоторых аспектах изобретения предусмотрены векторы или плазмиды (например, экспрессирующие векторы или упаковочные векторы), содержащие конструкции из нуклеиновой кислоты, включающие Cas9, подходящий для экспрессии в клетках хозяина, например, в клетках млекопитающих, дрожжевых клетках, клетках насекомых, клетках растений, грибковых клетках или клетках водорослей. В некоторых воплощениях конструкция из нуклеиновой кислоты содержит последовательность полинуклеотида, кодирующего Cas9, который экспрессируется в виде слитого белка с модифицированной транспозазой по изобретению.
Цинк-пальцевые белки
Настоящим изобретением также предусмотрены конструкции из нуклеиновой кислоты, содержащие полинуклеотиды, кодирующие цинк-пальцевые белки (ZFP) для встраивания экзогенной нуклеиновой кислоты в определенное место генома. В некоторых аспектах изобретения предусмотрены слитые белки, содержащие ZFP и модифицированную интегразу или модифицированную транспозазу по изобретению. В некоторых воплощениях изобретения предусмотрены нуклеиновые кислоты, кодирующие такие ZFP или слитые белки. В некоторых воплощениях изобретения предусмотрены плазмиды или экспрессирующие векторы, содержащие такие кодирующие нуклеиновые кислоты.
Используемые здесь цинк-пальцевые белки представляют собой белки, которые могут связываться с ДНК специфичным для последовательности образом. ZFP распределены неравномерно у эукариот. Идентифицированы такие ZFP, которые участвуют в распознавании ДНК, связывании РНК и связывании белков. Некоторые классификации цинк-пальцевых белков основываются на “группах складок” с учетом общей формы белкового остова в сложенном домене. Наиболее распространенными группами складок цинковых пальцев являются группы типа C2H2 или Cys2His2 (“классический цинковый палец”), скрипичного ключа и цинковой ленты. Репрезентативный мотив, характерный для одного класса этих белков (класса C2H2): -Cys-(X)2-4-Cys-(X)12-His-(X)3-5-His (где X – любая аминокислота).
ZFP по изобретению могут быть любые ZFP, их варианты или функциональные фрагменты, которые могут связываться с определенной последовательностью геномной ДНК в геноме. Неограничительные примеры ZFP включают ZFP, содержащие группы или мотивы складок цинковых пальцев, выбранные из доменов типа C2H2, gag-костяшек, скрипичного ключа, цинковой ленты, Zn2/Cys6 или TAZ2 и их комбинаций. В некоторых воплощениях ZFP представляют собой цинк-пальцевые белки типа C2H2. В некоторых воплощениях ZFP представляют собой сконструированные ZFP.
Для получения цинк-пальцевых нуклеаз можно слить сконструированные комплекты цинковых пальцев с ДНК-расщепляющим доменом (обычно с расщепляющим доменом FokI). Такие слияния цинковых пальцев и FokI стали полезными реагентами для манипулирования геномом.
ZFP по изобретению могут содержать 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12 и более доменов “цинковых пальцев”. ZFP могут содержать 2-12, 2-10, 2-8, 3-8, 4-8 или 5-8 доменов “цинковых пальцев”. В некоторых воплощениях ZFP содержат по 6 доменов “цинковых пальцев”.
Обычно процесс модульной сборки включает объединение отдельных цинковых пальцев, каждый из которых может распознавать последовательность ДНК из 3 пар оснований, получая комплекты из 3, 4, 5 или 6 пальцев, которые распознают целевые сайты длиной от 9 пар оснований до 18 пар оснований. В другом методе используют модули из 2 пальцев, получая комплекты цинковых пальцев из вплоть до 6 отдельных цинковых пальцев.
В некоторых воплощениях связывающий домен ZFP можно сконструировать так, чтобы он связывался с выбранной последовательностью. Сконструированный связывающий домен цинк-пальцевого белка может обладать лучшей специфичностью связывания по сравнению с природным ZFP. В некоторых воплощениях последовательность нуклеиновой кислоты, кодирующей ZFP, соответствует SEQ ID NO: 32, SEQ ID NO: 34, SEQ ID NO: 36 или SEQ ID NO: 38. В некоторых воплощениях аминокислотная последовательность ZFP соответствует к SEQ ID NO: 33, SEQ ID NO: 35, SEQ ID NO: 37 или SEQ ID NO: 39. В некоторых воплощениях ZFP имеет аминокислотную последовательность, которая по меньшей мере на 70%, на 75%, на 80%, на 85%, на 90%, на 95%, на 96%, на 97%, на 98%, на 99% или на 100% идентична последовательности любой из SEQ ID NO: 33, 35, 37 или 39.
В некоторых аспектах изобретения предусмотрены векторы или плазмиды (например, экспрессирующие векторы или упаковочные векторы), содержащие конструкции из нуклеиновой кислоты, включающие ZFP, подходящий для экспрессии в клетках хозяина, например, в клетках млекопитающих, дрожжевых клетках, клетках насекомых, клетках растений, грибковых клетках или клетках водорослей. В некоторых воплощениях конструкция из нуклеиновой кислоты содержит последовательность полинуклеотида, кодирующего ZFP, который экспрессируется в виде слитого белка с модифицированной интегразой или модифицированной транспозазой по изобретению.
VII Слитые белки
Настоящим изобретением предусмотрены слитые белки для сайт-специфического встраивания экзогенных нуклеиновых кислот в геном. В некоторых воплощениях слитый белок включает первый ДНК-связывающий белок, спроектированный для связывания с определенной последовательностью геномной ДНК, второй ДНК-связывающий белок, который обеспечивает встраивание экзогенной нуклеиновой кислоты в геном, причем второй ДНК-связывающий белок представляет собой интегразу или транспозазу по изобретению, и линкер, соединяющий первый и второй белок. В некоторых воплощениях первым ДНК-связывающим белком является белок Cas9 или цинк-пальцевый белок. В некоторых воплощениях первым ДНК-связывающим белком является Cas9, а вторым связывающим белком является описанная здесь модифицированная транспозаза, причем первый и второй связывающий белок могут располагаться в конструкции в любом порядке. В некоторых воплощениях первым ДНК-связывающим белком является цинк-пальцевый белок, а вторым связывающим белком является модифицированная интеграза, причем первый и второй связывающий белок могут располагаться в конструкции в любом порядке.
В некоторых воплощениях слитый белок включает линкер между первым связывающим белком и вторым связывающим белком, причем линкер содержит мотив (GGS)n, (GGGGS)n (SEQ ID NO: 133), (G)n, (EAAAK)n (SEQ ID NO: 134) или же на основе XTEN или (XP)n или любую комбинацию из них, где n независимо означает целое число от 1 до 50. В некоторых воплощениях линкер содержит от 12 до 24 аминокислот или же кодируется последовательностью нуклеиновой кислоты длиной от 36 до 72 нуклеотидов. В некоторых воплощениях линкер содержит последовательность XTEN либо последовательность GGS. В некоторых воплощениях слитый белок включает цинк-пальцевый белок, соединенный с модифицированной интегразой по изобретению, а линкер содержит последовательность GGS или последовательность XTEN, причем модифицированная интеграза может находиться с 5′- или 3′-стороны от линкера. В некоторых воплощениях слитый белок включает белок Cas9, соединенный с модифицированной транспозазой по изобретению, а линкер содержит последовательность GGS или же последовательность XTEN, причем модифицированная транспозаза может находиться с 5′- или 3′-стороны от линкера. В некоторых воплощениях линкером служит линкер, приведенный в таблице 1. В некоторых воплощениях линкер включает аминокислотную последовательность по SEQ ID NO: 49. В некоторых воплощениях линкер включает аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 49, SEQ ID NO: 51, SEQ ID NO: 53, SEQ ID NO: 55, SEQ ID NO: 57, SEQ ID NO: 59, SEQ ID NO: 61, SEQ ID NO: 63 либо их комбинаций. В некоторых воплощениях линкер кодируется последовательностью нуклеиновой кислоты, включающей SEQ ID NO: 48. В некоторых воплощениях линкер кодируется последовательностью нуклеиновой кислоты, включающей последовательность, выбранную из группы, состоящей из SEQ ID NO: 48, SEQ ID. NO: 50, SEQ ID NO: 52, SEQ ID NO: 54, SEQ ID NO: 56, SEQ ID NO: 58, SEQ ID NO: 60, SEQ ID NO: 62 либо их комбинаций.
Таблица 1. Линкеры
(SEQ ID NO)
(SEQ ID NO)
(SEQ ID NO: 48)
(SEQ ID NO: 50)
(SEQ ID NO: 55)
(SEQ ID NO: 57)
(SEQ ID NO: 58)
(SEQ ID NO: 59)
(SEQ ID NO: 62)
В некоторых воплощениях 3′-конец первого ДНК-связывающего белка соединяется с 5′-концом второго ДНК-связывающего белка при помощи линкера. В некоторых воплощениях 3′-конец второго ДНК-связывающего белка соединяется с 5′-концом первого ДНК-связывающего белка при помощи линкера. В некоторых воплощениях 3′-конец белка Cas9 соединяется с 5′-концом транспозазы при помощи линкера. В некоторых воплощениях 5′-конец белка Cas9 соединяется с 3′-концом транспозазы при помощи линкера. В некоторых воплощениях 3′-конец цинк-пальцевого белка соединяется с 5′-концом интегразы при помощи линкера. В некоторых воплощениях 5′-конец цинк-пальцевого белка соединяется с 3′-концом интегразы при помощи линкера.
При этом также предусмотрены слитые белки, полученные при экспрессии любых конструкций из нуклеиновой кислоты, предусмотренных в изобретении.
VIII Клетки/организмы хозяева
В некоторых воплощениях конструкции из нуклеиновой кислоты по изобретению экспрессируются в клетках-хозяевах. Подходящие клетки-хозяева включают, без ограничения, эукариотические и прокариотические клетки и/или линии клеток. Неограничительные примеры таких клеток-хозяев или клеточных линий, полученных из таких клеток, включают клетки COS, CHO (например, CHO-S, CHO-K1, CHO-DG44, CHO-DUXB11, CHO-DUKX, CHOK1SV), VERO, MDCK, WI38, V79, B14AF28-G3, BHK, HaK, NS0, SP2/0-Ag14, HeLa, HEK293 (например, HEK293-F, HEK293-H, HEK293-T) и perC6, а также клетки насекомых типа Spodoptera fugiperda (Sf) или грибковые клетки типа Saccharomyces, Pichia и Schizosaccharomyces.
В некоторых воплощениях клетки-хозяева происходят из микроорганизма. Микроорганизмы, которые применимы для некоторых способов, описанных здесь, включают, к примеру, бактерии (например, E. coli), дрожжевые клетки (например, Saccharomyces cerevisiae) и растения. Клетки-хозяева могут быть прокариотическими или эукариотическими. В некоторых воплощениях клетки-хозяева являются эукариотическими. Подходящие эукариотические клетки-хозяева включают, без ограничения, дрожжевые клетки, клетки насекомых, клетки растений, грибковые клетки и клетки водорослей.
В некоторых воплощениях клетками-хозяевами являются компетентные клетки. В некоторых воплощениях клетки-хозяева являются компетентными от природы. В некоторых воплощениях клетки-хозяева делаются компетентными, например, под действием процесса, в котором используется хлорид кальция и тепловой шок. Можно использовать любые компетентные клетки, особенно эукариотические клетки, в частности клетки млекопитающих, например, человека или животных. Это могут быть соматические, эмбриональные или стволовые клетки либо дифференцированные клетки. В некоторых аспектах такие клетки включают клетки 293T, клетки фибробластов, гепатоциты, мышечные клетки (скелетные, сердечные, гладкие, кровеносных сосудов и т.д.), нервные клетки (нейроны, глиальные клетки, астроциты) или эпителиальные клетки, почечные, глазные и т.д. Они также могут включать клетки насекомых, растений, дрожжевые либо прокариотические клетки. Кроме того, можно выделить первичные клетки и использовать их ex vivo для повторного введения подлежащему лечению субъекту после обработки нуклеазой (например, ZFN или TALEN) или системой нуклеаз (например, CRISPR/Cas). Подходящие первичные клетки включают мононуклеарные клетки периферической крови (PBMC) и другие субпопуляции клеток крови, как-то, без ограничения, T-лимфоциты типа T-клеток CD4+ или T-клеток CD8+. Подходящие клетки также включают и стволовые клетки, такие, к примеру, как эмбриональные стволовые клетки, индуцированные плюрипотентные стволовые клетки, гемопоэтические стволовые клетки (CD34+), нейрональные стволовые клетки и мезенхимальные стволовые клетки.
В некоторых воплощениях клетки-хозяева трансфицируют плазмидами, содержащими описанные здесь конструкции из нуклеиновой кислоты. В некоторых воплощениях плазмиды, содержащие конструкции из нуклеиновой кислоты, являются упаковочными плазмидами. В некоторых воплощениях плазмиды, содержащие конструкции из нуклеиновой кислоты, дополнительно содержат полинуклеотиды, кодирующие капсидные белки, например, gag и pol. В некоторых воплощениях клетки-хозяева трансфицируют (i) плазмидой, содержащей конструкцию из нуклеиновой кислоты, в комбинации с (ii) плазмидой, содержащей полинуклеотид, кодирующий белки оболочки вируса (плазмидой для оболочки); и (iii) плазмидой, содержащей последовательность экзогенной нуклеиновой кислоты (например, GOI), при этом вырабатываются вирусные частицы, содержащие экзогенную нуклеиновую кислоту, например, GOI, и слитый белок, включающий и первый, и второй связывающий белок.
В некоторых воплощениях клетки-хозяева трансфицируют (i) плазмидой, содержащей конструкцию из нуклеиновой кислоты, в комбинации с (ii) плазмидой, содержащей конструкцию из нуклеиновой кислоты, которая содержит полинуклеотид, кодирующий капсидные белки, например, gag и pol (это упаковочная плазмида, лишенная функциональной интегразы); (iii) плазмидой, содержащей полинуклеотид, кодирующий белки оболочки вируса (плазмида для оболочки); и (iv) плазмидой, содержащей последовательность экзогенной нуклеиновой кислоты (например, GOI), при этом вырабатываются вирусные частицы, содержащие экзогенную нуклеиновую кислоту, например, GOI, и слитый белок, включающий и первый, и второй связывающий белок.
В других воплощениях можно использовать вектор, например, лентивирусный вектор по изобретению, для доставки слитого белка, кодируемого конструкцией из нуклеиновой кислоты по изобретению, и экзогенной нуклеиновой кислоты в организм, например, млекопитающего, более предпочтительно в представляющие интерес целевые клетки млекопитающего. Лентивирусные векторы, содержащие слитые белки по изобретению, способны трансдуцировать различные типы клеток, как-то, к примеру, клетки печени (например, гепатоциты), мышечные клетки, клетки мозга, клетки почек, клетки сетчатки и гемопоэтические клетки. В некоторых воплощениях целевыми клетками настоящего изобретения являются “неделящиеся” клетки. Такие клетки включают клетки типа нервных клеток, которые обычно не делятся. Однако предполагается, что настоящее изобретение не ограничивается неделящимися клетками (включая, без ограничения, мышечные клетки, лейкоциты, клетки селезенки, клетки печени, глазные клетки, эпителиальные клетки и т.п.).
В некоторых воплощениях в организм вводятся упакованные слитые белки по изобретению, например, для редактирования генов в ДНК данного организма. В некоторых воплощениях таким организмом является человек. В некоторых воплощениях такими организмами являются другие млекопитающие. В некоторых воплощениях такими организмами являются другие приматы. В некоторых воплощениях такими организмами являются грызуны. В некоторых воплощениях такими организмами являются овцы, козы, крупный рогатый скот, кошки или собаки. В некоторых воплощениях такими организмами являются позвоночные, амфибии, рептилии, рыбы, насекомые, мухи или нематоды. В некоторых воплощениях такими организмами являются исследовательские животные. В некоторых воплощениях такие организмы являются генно-инженерными, например, это генно-инженерные субъекты, кроме человека. Такие организмы могут быть любого пола и на любой стадии развития.
IX Способ встраивания в геном
Были описаны методы встраивания экзогенных нуклеиновых кислот в геном. Например, см. Yusa et al., PNAS 4(108):1531-1536 (2011); Feng et al., Nuc. Acids Res. 4(38): 1204-1216 (2009); Kettlun et al., Amer. Soc. Gene and Cell Ther. 9(19):1636-1644 (2011); Skipper et al., 20(92):1-23 (2013); Li et al., PNAS 25: E2279-E2287 (2013); Mátés et al., Nature Genetics 41(6):753-761 (2009); Mali et al., Nat. Methods 10(10):957-963; Vargas et al., J. Trans. Med. 14(288):1-15 (2016); Gersbach et al., Acc. Chem. Res. 47:2309-2318 (2014); Chandrasegaran et al., Cell Gene Ther. Ins. 3(1):33-41 (2017); Wilson et al., 649:353-363 (2010); Zhao Zhang et al., Mol Ther Nucleic Acids 9: 230-241 (2017); Naldini L., EMBO Mol Med. 11(3) (2019); и Naldini L. et al., Hum Gene Ther. 27(10):727-728 (2016); которые все включены сюда путем ссылки.
Настоящим изобретением предусмотрены конструкции из нуклеиновой кислоты, кодирующие слитые белки для встраивания экзогенной нуклеиновой кислоты в определенное место генома. Настоящим изобретением также предусмотрены слитые белки для встраивания экзогенной нуклеиновой кислоты в определенное место генома. В некоторых воплощениях экзогенная нуклеиновая кислота для вставки может иметь длину до 5 т.п.н., до 10 т.п.н., до 15 т.п.н., 20 т.п.н., до 25 т.п.н., до 30 т.п.н., до 35 т.п.н. или до 40 т.п.н.
В другом воплощении предусмотрены способы сайт-специфического встраивания нуклеиновой кислоты в геном. В некоторых воплощениях способы включают контактирование целевой ДНК с каким-либо слитым белком, содержащим Cas9 и транспозазу, описанные здесь. Например, в некоторых воплощениях способ включает контактирование ДНК со слитым белком, содержащим два связанных полипептида: (i) Cas9; и (ii) транспозазу, причем активный Cas9 связывается с направляющей РНК, которая гибридизируется с участком ДНК, например, геномной ДНК.
В некоторых воплощениях способы включают контактирование целевой ДНК с каким-либо слитым белком, содержащим Cas9 и интегразу, описанные здесь. Например, в некоторых воплощениях способ включает контактирование ДНК со слитым белком, содержащим два связанных полипептида: (i) Cas9; и (ii) интегразу, причем активный Cas9 связывается с направляющей РНК, которая гибридизуется с участком ДНК, например, геномной ДНК.
В некоторых воплощениях способы включают контактирование целевой ДНК с каким-либо слитым белком, содержащим ZFP и интегразу, описанные здесь. Например, в некоторых воплощениях способ включает контактирование ДНК со слитым белком, содержащим два связанных полипептида: (i) ZFP; и (ii) интегразу, причем активный ZFP связывается с направляющей РНК, которая гибридизуется с участком ДНК, например, геномной ДНК.
В некоторых воплощениях слитый белок вводится в организм и/или клетки, содержащие целевую ДНК, например, геномную ДНК, с помощью вирусного вектора, например, лентивирусных частиц.
X Лентивирусная упаковка
Были описаны методы упаковки лентивирусов. См. Grandchamp et al., 9(6): 1-13 (2014); Voelkel et al., 107(17): 7805-7810 (2010); Tan et al., 80(4): 1939-1948; Li et al., 9(8):1-9 (2014); Mátés et al., Nature Genetics 41(6): 753-761 (2009); и Robert H Kutner et al., Nature Protocols 4(4): 495 (2009), которые все включены сюда путем ссылки.
Как правило, в лентивирусных системах доставки используется раздельная система с различными генами лентивируса на отдельных плазмидах, которая применяется для получения полного вируса, не содержащего генетических компонентов, необходимых для того, чтобы вызывать вирусные заболевания. Например, одна плазмида (плазмида для оболочки) может кодировать белки оболочки вируса (env); другая плазмида (упаковочная плазмида) может кодировать капсидные белки (например, gag и pol) и ферменты типа обратной транскриптазы и/или интегразы; а еще одна плазмида содержит представляющий интерес ген (GOI), фланкированный длинными концевыми повторами (для встраивания в геном), и последовательность psi (которая служит сигналом для упаковки гена в вирус) (плазмида для переноса). Если эти плазмиды ввести одновременно в клетку, то будут вырабатываться вирусы, содержащие GOI без вирусных генов, необходимых для того, чтобы вызывать заболевания.
В некоторых аспектах изобретения лентивирусные векторы (или частицы) по изобретению получают при помощи раздельной системы, например, системы транскомплементации (системы вектор/упаковка), путем трансфекции in vitro пермиссивных клеток (типа клеток 293T) плазмидой, содержащей определенные компоненты генома лентивирусного вектора, и еще по меньшей мере одной плазмидой, обеспечивающей in trans последовательности gag, pol и env, кодирующие полипептиды GAG, POL и белки оболочки или же часть этих полипептидов, достаточную для образования ретровирусных частиц.
Для примера, клетки хозяина трансфицируют a) упаковочной плазмидой, содержащей последовательность gag и pol лентивируса, b) второй плазмидой (плазмидой для оболочки или плазмидой для псевдотипирования env), содержащей ген, кодирующий белки оболочки (типа VSV-G), c) плазмидным вектором, содержащим последовательности 5′- и 3′-LTR, последовательность инкапсидации psi и трансген, и d) плазмидным вектором, содержащим конструкцию из нуклеиновой кислоты, кодирующую сконструированный слитый белок, описанный здесь. В некоторых воплощениях конструкция из нуклеиновой кислоты, кодирующая описанный здесь сконструированный слитый белок, находится на упаковочной плазмиде вместо отдельной плазмиды. Нуклеиновые кислоты, кодирующие кДНК gag, pol и env, можно легко получить стандартными методами из последовательностей вирусных генов, доступных на предшествующем уровне техники и в базах данных.
В некоторых воплощениях лентивирусный вектор содержит конструкцию из нуклеиновой кислоты, как описано здесь. В некоторых воплощениях лентивирусный вектор содержит слитый белок, как описано здесь.
В плазмидах используются промоторы, которые могут быть одинаковыми или разными. В некоторых воплощениях, в системе транскомплементации плазмид, плазмида для оболочки и плазмидный вектор, соответственно, для стимулирования экспрессии gag и pol или белков оболочки, мРНК из генома вектора и трансген содержат промоторы, которые могут быть одинаковыми или разными. Такие промоторы обычно выбирают из универсальных промоторов или специфичных, к примеру, из вирусных промоторов CMV, TK, LTR RSV и промоторов РНК-полимеразы III типа U6 или H1 или промоторов хелперных вирусов, кодирующих env, gag и pol (т.е. аденовирусных, бакуловирусных, герпесвирусных).
Для получения лентивирусных векторов по изобретению описанные здесь плазмиды можно ввести в клетки хозяина, продуцирующие вирусы, а затем собрать их. Подходящие клетки включают, без ограничения, эукариотические и прокариотические клетки и/или линии клеток. Неограничительные примеры таких клеток или клеточных линий, полученных из таких клеток, включают, например, клетки COS, CHO (например, CHO-S, CHO-K1, CHO-DG44, CHO-DUXB11, CHO-DUKX, CHOK1SV), VERO, MDCK, WI38, V79, B14AF28-G3, BHK, HaK, NS0, SP2/0-Ag14, HeLa, HEK293 (например, HEK293-F, HEK293-H, HEK293-T) и perC6, а также клетки насекомых типа Spodoptera fugiperda (Sf) или грибковые клетки типа Saccharomyces, Pichia и Schizosaccharomyces.
После трансфекции клеток хозяина плазмидами и получения лентивирусных векторов (или частиц) по изобретению можно очистить лентивирусные векторы (или частицы) по изобретению из супернатанта клеток. Очистка лентивирусных векторов для повышения их концентрации может осуществляться любым подходящим способом, как-то очисткой в градиенте плотности (например, хлорида цезия (CsCl)), методами хроматографии (например, колоночной или порционной хроматографии) или ультрацентрифугирования. Например, вектор по изобретению можно подвергнуть двум или трем стадиям очистки в градиенте плотности CsCl. Вектор предпочтительно очищают из трансфицированных клеток таким способом, который включает лизирование клеток, нанесение лизата на хроматографическую смолу, элюирование вируса из хроматографической смолы и сбор фракций, содержащих лентивирусный вектор по изобретению.
XI Способы доставки
Были описаны способы доставки лентивирусных векторов. Например, см. Vargas et al., J. Trans. Med. 14(288):1-15 (2016); Mali et al., Nat. Methods 10(10): 957-963; Mátés et al., Nature Genetics 41(6):753-761 (2009); Skipper et al., 20(92):1-23 (2013).
Лентивирусные векторы, содержащие слитые белки, кодируемые конструкциями из нуклеиновой кислоты по изобретению, можно вводить субъектам любым способом. В некоторых воплощениях лентивирусный вектор по изобретению вводится в клетки субъекта in vivo либо ex vivo.
В некоторых воплощениях лентивирусные векторы, содержащие слитые белки, кодируемые конструкциями из нуклеиновой кислоты по изобретению, применяются для доставки GOI и/или для воздействия на генетические дефекты ДНК у субъектов. В некоторых воплощениях лентивирусные векторы вводятся субъектам парентерально, предпочтительно интраваскулярно (в том числе внутривенно). При парентеральном введении векторы предпочтительно вводятся в фармацевтическом носителе, подходящем для инъекций, типа стерильного водного раствора или дисперсии.
В некоторых воплощениях лентивирусные векторы по изобретению применяются ex vivo.
В некоторых воплощениях лентивирусные векторы, содержащие слитые белки, кодируемые конструкциями из нуклеиновой кислоты по изобретению, применяются для доставки GOI и/или для воздействия на генетические дефекты ДНК у субъектов. В некоторых воплощениях у субъекта извлекают клетки и в них ex vivo вводят лентивирусный вектор, содержащий слитый белок, кодируемый конструкцией из нуклеиновой кислоты по изобретению, чтобы модифицировать ДНК этих клеток. Затем клетки, несущие модифицированную ДНК, размножают и вливают обратно субъекту. В некоторых воплощениях лентивирусные векторы, содержащие слитые белки, кодируемые конструкциями из нуклеиновой кислоты по изобретению, применяются для терапии T-клетками с химерным антигеновым рецептором (CAR), чтобы генетически модифицировать аутологичные T-клетки пациента для экспрессии CAR, специфичного к опухолевому антигену. В другом воплощении T-клетки с модифицированных CAR размножают ex vivo и вливают обратно пациенту. В некоторых воплощениях измененные T-клетки более специфически воздействуют на раковые клетки. В отличие от терапии антителами, T-клетки с CAR способны реплицироваться in vivo, что ведет к долгосрочной персистенции.
После введения лентивирусного вектора по изобретению или клеток, модифицированных ex vivo с помощью лентивирусного вектора по изобретению, за субъектом наблюдают для выявления экспрессии трансгена. Дозы и продолжительность лечения определяются индивидуально в зависимости от подлежащего лечению состояния или заболевания. Можно лечить различные состояния или заболевания, исходя из экспрессии гена, полученной при введения представляющего интерес гена в векторах по настоящему изобретению. Дозировка вектора при введении способом по изобретению будет варьироваться в зависимости от требуемой реакции организма и используемого вектора.
В некоторых применениях генной терапии желательно, чтобы вектор для генной терапии доставлялся с высокой степенью специфичности в определенный тип ткани. Соответственно, вирусный вектор можно модифицировать так, чтобы он обладал специфичностью к данному типу клеток, путем экспрессии лиганда в виде слитого белка с белком вирусной оболочки на внешней поверхности вируса. Лиганд выбирают так, чтобы он обладал сродством к рецептору, который присутствует в представляющем интерес типе клеток.
В некоторых аспектах изобретения предусмотрен способ встраивания последовательности экзогенной нуклеиновой кислоты в геномную ДНК организма, включающий: идентификацию определенной последовательности геномной ДНК в геноме организма; введение в организм лентивирусных частиц, содержащих конструкцию из нуклеиновой кислоты по изобретению, для связывания с определенной последовательностью геномной ДНК и встраивания экзогенной нуклеиновой кислоты в геномную ДНК; при этом экзогенная нуклеиновая кислота встраивается в данную последовательность геномной ДНК.
В некоторых аспектах изобретения предусмотрен способ контролируемого сайт-специфичного встраивания одной или нескольких копий последовательности экзогенной нуклеиновой кислоты в геном клеток, который включает: a) введение в клетки конструкции из нуклеиновой кислоты, вектора или слитого белка по изобретению и b) введение в клетки экзогенной нуклеиновой кислоты; при этом связывание слитого белка с определенной последовательностью геномной ДНК в геноме клеток приводит к расщеплению генома и встраиванию одной или нескольких копий экзогенной нуклеиновой кислоты в геном этих клеток. В некоторых аспектах введение в клетки осуществляется при помощи лентивирусных частиц.
XII Способы применения
Для проверки места встраивания и для отбора наилучших механизмов для направленного встраивания можно использовать несколько стратегий.
Для анализа описанной здесь модифицированной интегразы и транспозазы можно использовать репортерную линию клеток с промотором, половиной кодирующей последовательности GFP и донором сайта сплайсинга позади от сайта направленного встраивания в геном. Например, полезный груз лентивируса может содержать слитый вариант интегразы, за которым следует инвертированный сайт акцептора сплайсинга и другая половина GPF. Экспрессия GFP будет происходить тогда, когда непосредственно случится встраивание и при сплайсинге GFP-содержащей мРНК, образующейся из сайта вставки и встроенного полезного груза, образуется полная CDS GFP.
Для скрининга и сравнения мутантов по встраиванию также можно использовать системы транскомплементации VPR. Система транскомлементации может использоваться для направленного встраивания полезного груза лентивируса, содержащего слитый вариант интегразы, который при экспрессии и загрузке в частицы способствует встраиванию себя самого, будет загружена в вирусные частицы при слиянии с VPR. При этом будет восстанавливаться in trans встраивание дефектной IN, кодируемой в упаковочном векторе, используемом для получения частиц. Другие методы, которые можно использовать для картирования встраивания, включают зонды IC или FISH. Направленное встраивание также можно проверять по направленному разрушению TCRα или RFP либо активации GFP при направленном встраивании сайта сплайсинга.
При подходе FISH к совместному окрашиванию участка вставки и мишени в хроматине проводится флуоресцентная гибридизация in situ для локализации транспозона с GOI в геноме HEK293T. HEK293T можно трансфицировать 1) транспозоном GOI, 2) программируемой транспозазой и 3) направляющей РНК для PPP1R12. Разрабатывают зонды для посадки на ген PPP1R12, ген CD46 (в качестве отрицательного контроля) и GOI, которые можно синтезировать с помощью смеси Nick Translation Mix (Sigma) из ДНК, амплифицированной методом ПЦР.
В некоторых воплощениях слитый белок, содержащий модифицированную транспозазу или модифицированную интегразу, как описано здесь, улучшает специфичность встраивания экзогенной нуклеиновой кислоты в геном по сравнению со слитым белком, содержащим соответствующий белок дикого типа, например, при определении методом Genetrap. В некоторых воплощениях клетки HEK293T или любые другие пермиссивные клетки трансфицируют или трансдуцируют лентивирусными частицами со следующими плазмидами или полезными нагрузками: (i) плазмидой, содержащей направленную РНК, нацеленную на определенный участок ДНК, (ii) плазмидой, содержащей конструкцию из нуклеиновой кислоты по изобретению, кодирующую слитый белок модифицированной транспозазы или слитый белок модифицированной интегразы, и (iii) плазмидой Genetrap, содержащей последовательность нуклеиновой кислоты, кодирующей репортерный белок, например, GFP, лишенной промотора. В некоторых воплощениях плазмида Genetrap дополнительно содержит транспозон с инвертированными повторами.
В некоторых воплощениях можно определить процент клеток, содержащих вставку GFP, методом проточной цитометрии. В некоторых воплощениях слитый белок программируемой транспозазы повышает процент клеток, содержащих вставку GFP, по меньшей мере на 5%, на 10%, на 15%, на 20%, на 25% или на 30% по сравнению с соответствующим белком дикого типа. В некоторых воплощениях слитый белок программируемой транспозазы повышает процент клеток, содержащих вставку GFP, на 15-30%.
В некоторых воплощениях можно определить процент вставок в целевом сайте и степень охвата в целевом сайте (количество прочтений на 1 сайт вставки) путем экстракции геномной ДНК и направленного секвенирования с олигонуклеотидами, специфичными для вирусных LTR. В некоторых воплощениях слитый белок модифицированной транспозазы повышает процент вставок в целевом сайте по меньшей мере в 10 раз, в 20 раз, в 30 раз, в 40 раз, в 50 раз, в 60 раз, в 70 раз, в 80 раз, в 90 раз или в 100 раз по сравнению с соответствующим белком дикого типа. В некоторых воплощениях процент вставок в целевом сайте повышается в 10-100 раз. В некоторых воплощениях слитый белок модифицированной транспозазы повышает степень охвата в целевом сайте (количество прочтений на 1 сайт вставки) по меньшей мере в 10 раз, в 20 раз, в 30 раз, в 40 раз, в 50 раз, в 60 раз, в 70 раз, в 80 раз, в 90 раз, в 100 раз, в 110 раз, в 120 раз, в 130 раз, в 140 раз, в 150 раз, в 160 раз, в 170 раз, в 180 раз, в 190 раз или в 200 раз по сравнению с соответствующим белком дикого типа. В некоторых воплощениях степень охвата в целевом сайте (количество прочтений на 1 сайт вставки) повышается по меньшей мере в 100 раз.
В некоторых воплощениях слитый белок модифицированной интегразы улучшает специфичность встраивания экзогенной нуклеиновой кислоты в геном по сравнению с соответствующим белком дикого типа при определении по встраиванию GFP. В некоторых воплощениях получают лентивирус, содержащий слитый белок модифицированной интегразы, путем трансфекции клеток HEK293T или любых других пермиссивных клеток (i) плазмидой, содержащей последовательность нуклеиновой кислоты, кодирующей GFP, (ii) плазмидой, содержащей упаковочные белки, (iii) плазмидой, содержащей белок оболочки, и (iv) плазмидой, содержащей конструкцию из нуклеиновой кислоты, кодирующую слитый белок модифицированной интегразы. Супернатант, содержащий лентивирус, собирали через 48 ч после трансфекции.
Для направленного встраивания клетки HEK293T трансфицировали лентивирусом, содержащим слитый белок модифицированной интегразы. В некоторых воплощениях определяли процент GFP-положительных клеток методом проточной цитометрии через 3, 5, 7, 10 и 12 дней после трансфекции. В некоторых воплощениях слитый белок модифицированной интегразы повышает процент клеток, содержащих вставку GFP, по меньшей мере на 5%, на 10%, на 15%, на 20%, на 25% или на 30%. по сравнению с соответствующим белком дикого типа.
В некоторых воплощениях можно определить процент вставок в целевом сайте и степень охвата в целевом сайте (количество прочтений на 1 сайт вставки) путем экстракции геномной ДНК и направленного секвенирования с олигонуклеотидами, специфичными для вставленных вирусных LTR. В некоторых воплощениях слитый белок модифицированной интегразы повышает процент вставок в целевом сайте по меньшей мере в 10 раз, в 20 раз, в 30 раз, в 40 раз, в 50 раз, в 60 раз, в 70 раз, в 80 раз, в 90 раз или в 100 раз по сравнению с соответствующим белком дикого типа. В некоторых воплощениях слитый белок модифицированной интегразы повышает степень охвата в целевом сайте (количество прочтений на 1 сайт вставки) по меньшей мере в 10 раз, в 20 раз, в 30 раз, в 40 раз, в 50 раз, в 60 раз, в 70 раз, в 80 раз, в 90 раз, в 100 раз, в 110 раз, в 120 раз, в 130 раз, в 140 раз, в 150 раз, в 160 раз, в 170 раз, в 180 раз, в 190 раз или в 200 раз по сравнению с соответствующим белком дикого типа.
Возможные применения лентивирусных векторов, содержащих слитые белки по изобретению, включают генную терапию, то есть перенос генов в любые клетки млекопитающих, в частности, в клетки человека. Это могут быть делящиеся клетки или покоящиеся клетки, клетки, принадлежащие центральным органам или периферическим органам, таким как печень, поджелудочная железа, мышцы, сердце и т.п. Генная терапия может обеспечить экспрессию белков, например, нейротрофических факторов, ферментов, факторов транскрипции, рецепторов и т.п. Лентивирусные векторы по изобретению также могут особенно подходить для исследовательских целей.
В некоторых воплощениях конструкции из нуклеиновой кислоты, слитые белки и/или лентивирусные векторы по изобретению вводятся субъектам для лечения заболеваний. В некоторых воплощениях заболевания представляют собой генетические заболевания, при которых может принести пользу генная терапия.
В некоторых воплощениях лентивирусные векторы, содержащие слитые белки по изобретению, могут применяться в качестве лекарственных средств. Лентивирусные векторы по изобретению могут особенно подходить для лечения генетических заболеваний у субъектов.
XIII Композиции и наборы
Настоящим изобретением также предусмотрены композиции для осуществления описанных здесь способов на практике. В некоторых воплощениях композиции содержат конструкцию из нуклеиновой кислоты или вектор, как определено в изобретении, и последовательность полинуклеотида, кодирующего экзогенную нуклеиновую кислоту для вставки в геном, которая содержится в упаковочном векторе или связана с ним.
В некоторых воплощениях конструкция из нуклеиновой кислоты находится в виде РНК, ДНК или белка, а последовательность полинуклеотида, кодирующего экзогенную нуклеиновую кислоту, находится в виде РНК или ДНК, в зависимости от способа доставки. Предпочтительно последовательность полинуклеотида, кодирующего экзогенную нуклеиновую кислоту, находится в виде РНК.
В некоторых воплощениях композиции не содержат вирусов, а упаковочный вектор представляет собой наночастицы, например, полимерные или липидные наночастицы. Упаковочным вектором также может быть носитель, связанный с элементами композиции. В некоторых воплощениях композиции содержатся в вирусных векторах, предпочтительно в лентивирусных частицах.
В некоторых воплощениях композиция включает (a) конструкцию из нуклеиновой кислоты, описанную здесь (например, содержащую Cas9 и транспозазу), в виде РНК, (b) направляющую РНК, если она нужна (например, в виде отдельной линейной одноцепочечной молекулы РНК), и (c) полинуклеотид, содержащий экзогенный ген для вставки в виде ДНК (например, в векторе), который содержится в упаковочном векторе или связан с ним.
В некоторых воплощениях композиция включает (a) слитый белок, описанный здесь (например, содержащий Cas9 и транспозазу), в виде белка, (b) направляющую РНК, если она нужна (например, в виде отдельной линейной одноцепочечной молекулы РНК), причем слитый белок и направляющая РНК образуют рибонуклеиново-белковый комплекс (RNP), и (c) полинуклеотид, содержащий экзогенный ген для вставки в виде ДНК (например, в векторе), который содержится в упаковочном векторе или связан с ним.
В некоторых воплощениях композиция включает (a) конструкцию из нуклеиновой кислоты, описанную здесь (например, содержащую Cas9 и транспозазу), в виде ДНК, (b) направляющую РНК, если она нужна (например, в виде отдельной линейной молекулы РНК или в виде ДНК в векторе), и (c) полинуклеотид, содержащий экзогенный ген для вставки в виде ДНК (например, в векторе), который содержится в упаковочном векторе или связан с ним.
В некоторых воплощениях композиция включает (a) слитый белок, описанный здесь (например, содержащий Cas9 и интегразу), в виде белка, (b) направляющую РНК, если она нужна (например, в виде отдельной молекулы РНК, образующей комплекс со слитым белком), и (c) полинуклеотид, содержащий экзогенный ген для вставки, который содержится в упаковочном векторе или связан с ним. В предпочтительном воплощении упаковочный вектор представляет собой лентивирусные частицы. В некоторых воплощениях слитый белок (a) связан с капсидом лентивируса при помощи gag-pol или VPR (вирусного белка R). В некоторых воплощениях полинуклеотид (c) находится в виде РНК в качестве полезного груза интегразы.
В предпочтительном воплощении при использовании ZFP гидовая РНК (b) может не понадобиться.
Настоящим изобретением также предусмотрены наборы для осуществления описанных здесь способов на практике. Наборы могут содержать конструкции из нуклеиновых кислот или слитые белки, как описано здесь. В некоторых аспектах наборы могут содержать лентивирусные частицы, содержащие конструкции из нуклеиновой кислоты или слитые белки, как описано здесь.
Данные наборы также могут содержать инструкции по применению компонентов набора для осуществления данных способов на практике. Инструкции по осуществлению данных способов на практике обычно записываются на подходящий носитель для записи. Например, инструкции могут быть напечатаны на подложке типа бумаги или пластика и т.п. При этом инструкции могут находиться в наборе в виде упаковочного вкладыша, на этикетке контейнера из набора или его компонентов (т.е. связанными с упаковкой или субупаковкой) и т.п. В других воплощениях инструкции находятся в виде файла данных для электронного хранения на подходящем для компьютера носителе данных, например, CD-ROM, дискеты и т.п. Еще в других воплощениях фактически инструкций нет в наборе, но предусмотрены средства для получения инструкций из удаленного источника, например, через Интернет. Примером этого воплощения является набор, содержащий веб-адрес, по которому инструкции можно просмотреть и/или с которого их можно загрузить. Как и сами инструкции, это средство получения инструкций записано на подходящей подложке.
XIV Воплощения
E1. Конструкция из нуклеиновой кислоты, включающая:
a) последовательность первого полинуклеотида, кодирующего первый ДНК-связывающий белок, сконструированный для связывания с определенной последовательностью геномной ДНК в геноме;
b) последовательность второго полинуклеотида, кодирующего второй ДНК-связывающий белок, который обеспечивает встраивание экзогенной нуклеиновой кислоты в геном, причем второй ДНК-связывающий белок представляет собой (i) интегразу, модифицированную относительно интегразы дикого типа, или (ii) транспозазу, модифицированную относительно транспозазы дикого типа; и
c) последовательность третьего полинуклеотида, включающего нуклеиновую кислоту, кодирующую линкер;
причем конструкция из нуклеиновой кислоты кодирует слитый белок, включающий первый ДНК-связывающий белок, второй ДНК-связывающий белок и линкер между первым ДНК-связывающим белком и вторым ДНК-связывающим белком.
E2. Конструкция из нуклеиновой кислоты по воплощению E1, при этом второй ДНК-связывающий белок модифицирован для повышения специфичности встраивания экзогенной нуклеиновой кислоты в геном по сравнению с соответствующим белком дикого типа.
E3. Конструкция из нуклеиновой кислоты по воплощению E1 или E2, при этом экзогенная нуклеиновая кислота для вставки может иметь длину до 20 т.п.н.
E4. Конструкция из нуклеиновой кислоты по любому из воплощений E1-E3, при этом последовательность первого полинуклеотида кодирует белок, выбранный из группы, состоящей из цинк-пальцевых белков, белка Cas9 и их вариантов или функциональных фрагментов.
E5. Конструкция из нуклеиновой кислоты по воплощению E4, при этом белок Cas9 выбран из группы, состоящей из Cas9 человека, никазы Cas9, Cas9 Streptococcus pyogenes, Cas9 Staphylococcus aureus, Cas12a, Cas12b и «мертвого» Cas9.
E6. Конструкция из нуклеиновой кислоты по воплощению E4, при этом цинк-пальцевый белок представляет собой цинк-пальцевый белок типа C2H2.
E7. Конструкция из нуклеиновой кислоты по любому из воплощений E1-E6, при этом модифицированная интеграза представляет собой модифицированную интегразу вируса иммунодефицита человека (ВИЧ) или её функциональный фрагмент.
E8. Конструкция из нуклеиновой кислоты по воплощению E7, при этом модифицированная интеграза ВИЧ содержит мутации по одной или нескольким из числа аминокислот 10, 13, 64, 94, 116, 117, 119, 120, 122, 124, 128, 152, 168, 170, 185, 231, 264, 266 или 273 в соответствии с нумерацией аминокислот в последовательности интегразы ВИЧ дикого типа (SEQ ID NO: 1).
E9. Конструкция из нуклеиновой кислоты по воплощению E8, при этом мутации модифицированной интегразы ВИЧ включают одну или несколько из мутаций D10K, E13K, D64A, D64E, G94D, G94E, G94R, G94K, D116A, D116E, N117D, N117E, N117R, N117K, S119A, S119P, S119T, S119G, S119D, S119E, S119R, S119K, N120D, N120E, N120R, N120K, T122K, T122I, T122V, T122A, T122R, A124D, A124E, A124R, A124K, A128T, E152A, E152D, Q168L, Q168A, E170G, F185K, R231G, R231K, R231D, R231E, R231S, K264R, K266R или K273R в соответствии с нумерацией аминокислот в последовательности интегразы ВИЧ дикого типа (SEQ ID NO: 1).
E10. Конструкция из нуклеиновой кислоты по любому из воплощений E7-E9, при этом модифицированная интеграза ВИЧ имеет аминокислотную последовательность, которая по меньшей мере на 85%, на 90% или на 95% идентична последовательности, приведенной в SEQ ID NO: 3.
E11. Конструкция из нуклеиновой кислоты по любому из воплощений E1-E6, при этом модифицированная транспозаза выбрана из группы, состоящей из модифицированной Frog Prince, модифицированной Sleeping Beauty, модифицированной гиперактивной Sleeping Beauty (SB100X), модифицированной PiggyBac, модифицированной гиперактивной PiggyBac и их функциональных фрагментов.
E12. Конструкция из нуклеиновой кислоты по воплощению E11, при этом модифицированная транспозаза представляет собой модифицированную гиперактивную PiggyBac или её функциональный фрагмент.
E13. Конструкция из нуклеиновой кислоты по воплощению E12, при этом модифицированная гиперактивная PiggyBac содержит мутации по одной или нескольким из числа аминокислот 245, 268, 275, 277, 287, 290, 315, 325, 341, 346, 347, 350, 351, 356, 357, 372, 375, 388, 409, 412, 432, 447, 450, 460, 461, 465, 517, 560, 564, 571, 573, 576, 586, 587, 589, 592 и 594 в соответствии с нумерацией аминокислот в последовательности гиперактивной PiggyBac (SEQ ID NO: 9).
E14. Конструкция из нуклеиновой кислоты по воплощению E13, при этом мутации модифицированной гиперактивной PiggyBac включают одну или несколько из мутаций R245A, D268N, R275A/R277A, K287A, K290A, K287A/K290A, R315A, G325A, R341A, D346N, N347A, N347S, T350A, S351E, S351P, S351A, K356E, N357A, R372A, K375A, R372A/K375A, R388A, K409A, K412A, K409A/K412A, K432A, D447A, D447N, D450N, R460A, K461A, R460A/K461A, W465A, S517A, T560A, S564P, S571N, S573A, K576A, H586A, I587A, M589V, S592G или F594L в соответствии с нумерацией аминокислот в последовательности гиперактивной PiggyBac (SEQ ID NO: 9).
E15. Конструкция из нуклеиновой кислоты по любому из воплощений E12-E14, при этом модифицированная гиперактивная PiggyBac имеет аминокислотную последовательность, которая по меньшей мере на 85%, на 90% или на 95% идентична последовательности, приведенной в SEQ ID NO: 10.
E16. Конструкция из нуклеиновой кислоты по любому из воплощений E1-E15, при этом линкер включает последовательность XTEN или последовательность GGS.
E17. Конструкция из нуклеиновой кислоты по любому из воплощений E1-E16, при этом последовательность, кодирующая линкер, имеет длину от 9 до 150 нуклеотидов.
E18. Конструкция из нуклеиновой кислоты по любому из воплощений E1-E17, при этом 3′-конец последовательности первого полинуклеотида соединяется с 5′-концом второго полинуклеотида линкером из нуклеиновой кислоты.
E19. Конструкция из нуклеиновой кислоты по любому из воплощений E1-E17, при этом 3′-конец последовательности второго полинуклеотида соединяется с 5′-концом последовательности первого полинуклеотида линкером из нуклеиновой кислоты.
E20. Вектор, содержащий конструкцию из нуклеиновой кислоты по любому из воплощений E1-E19, при этом экспрессирующий вектор подходит для экспрессии в клетках млекопитающих, дрожжевых клетках, клетках насекомых, клетках растений, грибковых клетках или клетках водорослей.
E21. Конструкция из нуклеиновой кислоты по воплощению E1, при этом:
a) последовательность первого полинуклеотида кодирует белок Cas9; а
b) последовательность второго полинуклеотида кодирует модифицированную транспозазу, а именно модифицированную гиперактивную PiggyBac или её функциональный фрагмент.
E22. Конструкция из нуклеиновой кислоты по воплощению E21, при этом белок Cas9 выбран из группы, состоящей из Cas9 человека, никазы Cas9, Cas9 Streptococcus pyogenes, Cas9 Staphylococcus aureus, Cas12a, Cas12b и «мертвого» Cas9.
E23. Конструкция из нуклеиновой кислоты по воплощению E21 или E22, при этом модифицированная гиперактивная PiggyBac содержит мутации по одной или нескольким из числа аминокислот 245, 268, 275, 277, 287, 290, 315, 325, 341, 346, 347, 350, 351, 356, 357, 372, 375, 388, 409, 412, 432, 447, 450, 460, 461, 465, 517, 560, 564, 571, 573, 576, 586, 587, 589, 592 и 594 в соответствии с нумерацией аминокислот в последовательности гиперактивной PiggyBac (SEQ ID NO: 9).
E24. Конструкция из нуклеиновой кислоты по воплощению E23, при этом мутации модифицированной гиперактивной PiggyBac включают одну или несколько из мутаций R245A, D268N, R275A/R277A, K287A, K290A, K287A/K290A, R315A, G325A, R341A, D346N, N347A, N347S, T350A, S351E, S351P, S351A, K356E, N357A, R372A, K375A, R372A/K375A, R388A, K409A, K412A, K409A/K412A, K432A, D447A, D447N, D450N, R460A, K461A, R460A/K461A, W465A, S517A, T560A, S564P, S571N, S573A, K576A, H586A, I587A, M589V, S592G или F594L в соответствии с нумерацией аминокислот в последовательности гиперактивной PiggyBac (SEQ ID NO: 9).
E25. Конструкция из нуклеиновой кислоты по воплощению E21 или E22, при этом модифицированная гиперактивная PiggyBac имеет аминокислотную последовательность, которая по меньшей мере на 85%, на 90% или на 95% идентична последовательности, приведенной в SEQ ID NO: 10.
E26. Конструкция из нуклеиновой кислоты по любому из воплощений E21-E25, при этом линкер включает последовательность XTEN или последовательность GGS.
E27. Конструкция из нуклеиновой кислоты по любому из воплощений E21-E26, при этом последовательность, кодирующая линкер, имеет длину от 9 до 150 нуклеотидов.
E28. Конструкция из нуклеиновой кислоты по любому из воплощений E22-E27, при этом 3′-конец последовательности второго полинуклеотида соединяется с 5′-концом последовательности первого полинуклеотида при помощи линкера.
E29. Конструкция из нуклеиновой кислоты по воплощению E1, при этом:
a) последовательность первого полинуклеотида кодирует цинк-пальцевый белок; а
b) последовательность второго полинуклеотида кодирует модифицированную интегразу или её функциональный фрагмент.
E30. Конструкция из нуклеиновой кислоты по воплощению E29, при этом цинк-пальцевый белок представляет собой цинк-пальцевый белок типа C2H2.
E31. Конструкция из нуклеиновой кислоты по воплощению E29 или E30, при этом модифицированная интеграза представляет собой модифицированную интегразу вируса иммунодефицита человека (ВИЧ) или её функциональный фрагмент.
E32. Конструкция из нуклеиновой кислоты по воплощению E31, при этом модифицированная интеграза ВИЧ содержит мутации по одной или нескольким из числа аминокислот 10, 13, 64, 94, 116, 117, 119, 120, 122, 124, 128, 152, 168, 170, 185, 231, 264, 266 или 273 в соответствии с нумерацией аминокислот в последовательности интегразы ВИЧ дикого типа (SEQ ID NO: 1).
E33. Конструкция из нуклеиновой кислоты по воплощению E32, при этом мутации модифицированной интегразы ВИЧ включают одну или несколько из мутаций D10K, E13K, D64A, D64E, G94D, G94E, G94R, G94K, D116A, D116E, N117D, N117E, N117R, N117K, S119A, S119P, S119T, S119G, S119D, S119E, S119R, S119K, N120D, N120E, N120R, N120K, T122K, T122I, T122V, T122A, T122R, A124D, A124E, A124R, A124K, A128T, E152A, E152D, Q168L, Q168A, E170G, F185K, R231G, R231K, R231D, R231E, R231S, K264R, K266R или K273R в соответствии с нумерацией аминокислот в последовательности интегразы ВИЧ дикого типа (SEQ ID NO: 1).
E34. Конструкция из нуклеиновой кислоты по любому из воплощений E31-E33, при этом модифицированная интеграза ВИЧ имеет аминокислотную последовательность, которая по меньшей мере на 85%, на 90% или на 95% идентична последовательности, приведенной в SEQ ID NO: 3.
E35. Конструкция из нуклеиновой кислоты по любому из воплощений E29-E34, при этом включает последовательность XTEN или последовательность GGS.
E36. Конструкция из нуклеиновой кислоты по любому из воплощений E29-E35, при этом последовательность, кодирующая линкер, имеет длину от 9 до 150 нуклеотидов.
E37. Конструкция из нуклеиновой кислоты по любому из воплощений E29-E36, при этом 3′-конец последовательности второго полинуклеотида соединяется с 5′-концом последовательности первого полинуклеотида при помощи линкера.
E38. Вектор, содержащий конструкцию из нуклеиновой кислоты по любому из воплощений E21-E37, при этом экспрессирующий вектор подходит для экспрессии в клетках млекопитающих, дрожжевых клетках, клетках насекомых, клетках растений, грибковых клетках или клетках водорослей.
E39. Клетки-хозяева, содержащие конструкцию из нуклеиновой кислоты или вектор по любому из воплощений E1-E38.
E40. Слитый белок, включающий:
первый ДНК-связывающий белок, сконструированный для связывания с определенной последовательностью геномной ДНК в геноме;
второй ДНК-связывающий белок, который обеспечивает встраивание экзогенной нуклеиновой кислоты в геном, причем второй ДНК-связывающий белок представляет собой интегразу или транспозазу, модифицированную относительно дикого типа; и
линкер, соединяющий первый белок и второй белок.
E41. Слитый белок по воплощению E40, при этом второй ДНК-связывающий белок модифицирован для повышения специфичности встраивания экзогенной нуклеиновой кислоты в геном по сравнению с соответствующим белком дикого типа.
E42. Слитый белок по воплощению E40 или E41, при этом экзогенная нуклеиновая кислота может иметь длину до 20 т.п.н.
E43. Слитый белок по любому из воплощений E40-E42, при этом первый ДНК-связывающий белок выбран из группы, состоящей из цинк-пальцевых белков, белка Cas9 и их вариантов или функциональных фрагментов.
E44. Слитый белок по воплощению E43, при этом белок Cas9 выбран из группы, состоящей из Cas9 человека, никазы Cas9, Cas9 Streptococcus pyogenes, Cas9 Staphylococcus aureus, Cas12a, Cas12b и «мертвого» Cas9.
E45. Слитый белок по воплощению E43, при этом цинк-пальцевый белок представляет собой цинк-пальцевый белок типа C2H2.
E46. Слитый белок по любому из воплощений E40-E45, при этом модифицированная интеграза представляет собой модифицированную интегразу вируса иммунодефицита человека (ВИЧ) или её функциональный фрагмент.
E47. Слитый белок по воплощению E46, при этом модифицированная интеграза ВИЧ содержит мутации по одной или нескольким из числа аминокислот 10, 13, 64, 94, 116, 117, 119, 120, 122, 124, 128, 152, 168, 170, 185, 231, 264, 266 или 273 в соответствии с нумерацией аминокислот в последовательности интегразы ВИЧ дикого типа (SEQ ID NO: 1).
E48. Слитый белок по воплощению E47, при этом мутации модифицированной интегразы ВИЧ включают одну или несколько из мутаций D10K, E13K, D64A, D64E, G94D, G94E, G94R, G94K, D116A, D116E, N117D, N117E, N117R, N117K, S119A, S119P, S119T, S119G, S119D, S119E, S119R, S119K, N120D, N120E, N120R, N120K, T122K, T122I, T122V, T122A, T122R, A124D, A124E, A124R, A124K, A128T, E152A, E152D, Q168L, Q168A, E170G, F185K, R231G, R231K, R231D, R231E, R231S, K264R, K266R или K273R в соответствии с нумерацией аминокислот в последовательности интегразы ВИЧ дикого типа (SEQ ID NO: 1).
E49. Слитый белок по любому из воплощений E46-E48, при этом модифицированная интеграза ВИЧ имеет аминокислотную последовательность, которая по меньшей мере на 85%, на 90% или на 95% идентична последовательности, приведенной в SEQ ID NO: 3.
E50. Слитый белок по любому из воплощений E40-E45, при этом модифицированная транспозаза выбрана из группы, состоящей из модифицированной Frog Prince, модифицированной Sleeping Beauty, модифицированной гиперактивной Sleeping Beauty (SB100X), модифицированной PiggyBac, модифицированной гиперактивной PiggyBac и их функциональных фрагментов.
E51. Слитый белок по воплощению E50, при этом модифицированная транспозаза представляет собой модифицированную гиперактивную PiggyBac или её функциональный фрагмент.
E52. Слитый белок по воплощению E51, при этом модифицированная гиперактивная PiggyBac содержит мутации по одной или нескольким из числа аминокислот 245, 268, 275, 277, 287, 290, 315, 325, 341, 346, 347, 350, 351, 356, 357, 372, 375, 388, 409, 412, 432, 447, 450, 460, 461, 465, 517, 560, 564, 571, 573, 576, 586, 587, 589, 592 и 594 в соответствии с нумерацией аминокислот в последовательности гиперактивной PiggyBac (SEQ ID NO: 9).
E53. Слитый белок по воплощению E52, при этом мутации модифицированной гиперактивной PiggyBac включают одну или несколько из мутаций R245A, D268N, R275A/R277A, K287A, K290A, K287A/K290A, R315A, G325A, R341A, D346N, N347A, N347S, T350A, S351E, S351P, S351A, K356E, N357A, R372A, K375A, R372A/K375A, R388A, K409A, K412A, K409A/K412A, K432A, D447A, D447N, D450N, R460A, K461A, R460A/K461A, W465A, S517A, T560A, S564P, S571N, S573A, K576A, H586A, I587A, M589V, S592G или F594L в соответствии с нумерацией аминокислот в последовательности гиперактивной PiggyBac (SEQ ID NO: 9).
E54. Слитый белок по любому из воплощений E50-E53, при этом модифицированная гиперактивная PiggyBac имеет аминокислотную последовательность, которая по меньшей мере на 85%, на 90% или на 95% идентична последовательности, приведенной в SEQ ID NO: 10.
E55. Слитый белок по любому из воплощений E40-E54, при этом линкер включает последовательность XTEN или последовательность GGS.
E56. Слитый белок по любому из воплощений E40-E55, при этом линкер имеет длину от 3 до 50 аминокислот.
E57. Слитый белок по воплощению E40, при этом:
a) первый ДНК-связывающий белок представляет собой белок Cas9; а
b) второй ДНК-связывающий белок представляет собой модифицированную гиперактивную PiggyBac или её функциональный фрагмент.
E58. Слитый белок по воплощению E57, при этом белок Cas9 выбран из группы, состоящей из Cas9 человека, никазы Cas9, Cas9 Streptococcus pyogenes, Cas9 Staphylococcus aureus, Cas12a, Cas12b и «мертвого» Cas9.
E59. Слитый белок по воплощению E57 или E58, при этом модифицированная гиперактивная PiggyBac содержит мутации по одной или нескольким из числа аминокислот 245, 268, 275, 277, 287, 290, 315, 325, 341, 346, 347, 350, 351, 356, 357, 372, 375, 388, 409, 412, 432, 447, 450, 460, 461, 465, 517, 560, 564, 571, 573, 576, 586, 587, 589, 592 и 594 в соответствии с нумерацией аминокислот в последовательности гиперактивной PiggyBac (SEQ ID NO: 9).
E60. Слитый белок по воплощению E59, при этом мутации модифицированной гиперактивной PiggyBac включают одну или несколько из мутаций R245A, D268N, R275A/R277A, K287A, K290A, K287A/K290A, R315A, G325A, R341A, D346N, N347A, N347S, T350A, S351E, S351P, S351A, K356E, N357A, R372A, K375A, R372A/K375A, R388A, K409A, K412A, K409A/K412A, K432A, D447A, D447N, D450N, R460A, K461A, R460A/K461A, W465A, S517A, T560A, S564P, S571N, S573A, K576A, H586A, I587A, M589V, S592G или F594L в соответствии с нумерацией аминокислот в последовательности гиперактивной PiggyBac (SEQ ID NO: 9).
E61. Слитый белок по любому из воплощений E57-E60, при этом модифицированная гиперактивная PiggyBac имеет аминокислотную последовательность, которая по меньшей мере на 85%, на 90% или на 95% идентична последовательности, приведенной в SEQ ID NO: 10.
E62. Слитый белок по воплощению E40, при этом:
a) первый ДНК-связывающий белок представляет собой цинк-пальцевый белок; а
b) второй ДНК-связывающий белок представляет собой модифицированную интегразу или её функциональный фрагмент.
E63. Слитый белок по воплощению E62, при этом цинк-пальцевый белок представляет собой цинк-пальцевый белок типа C2H2.
E64. Слитый белок по воплощению E62 или E63, при этом модифицированная интеграза представляет собой модифицированную интегразу вируса иммунодефицита человека (ВИЧ) или её функциональный фрагмент.
E65. Слитый белок по воплощению E64, при этом модифицированная интеграза ВИЧ содержит мутации по одной или нескольким из числа аминокислот 10, 13, 64, 94, 116, 117, 119, 120, 122, 124, 128, 152, 168, 170, 185, 231, 264, 266 или 273 в соответствии с нумерацией аминокислот в последовательности интегразы ВИЧ дикого типа (SEQ ID NO: 1).
E66. Слитый белок по воплощению E65, при этом мутации модифицированной интегразы ВИЧ включают одну или несколько из мутаций D10K, E13K, D64A, D64E, G94D, G94E, G94R, G94K, D116A, D116E, N117D, N117E, N117R, N117K, S119A, S119P, S119T, S119G, S119D, S119E, S119R, S119K, N120D, N120E, N120R, N120K, T122K, T122I, T122V, T122A, T122R, A124D, A124E, A124R, A124K, A128T, E152A, E152D, Q168L, Q168A, E170G, F185K, R231G, R231K, R231D, R231E, R231S, K264R, K266R или K273R в соответствии с нумерацией аминокислот в последовательности интегразы ВИЧ дикого типа (SEQ ID NO: 1).
E67. Слитый белок по воплощению E62, при этом модифицированная интеграза ВИЧ имеет аминокислотную последовательность, которая по меньшей мере на 85%, на 90% или на 95% идентична последовательности, приведенной в SEQ ID NO: 3.
E68. Слитый белок по любому из воплощений E57-E67, при этом линкер включает последовательность XTEN или последовательность GGS.
E69. Слитый белок по любому из воплощений E57-E68, при этом линкер имеет длину от 3 до 50 аминокислот.
E70. Слитый белок по любому из воплощений E40-E69, при этом 3′-конец второго ДНК-связывающего белка соединяется с 5′-концом первого ДНК-связывающего белка при помощи линкера.
E71. Лентивирусные частицы, содержащие слитый белок по любому из воплощений E40-E69.
E72. Способ получения лентивирусных частиц для редактирования генов, включающий экспрессирование в клетках хозяина:
a) полинуклеотида, содержащего конструкцию из нуклеиновой кислоты по любому из воплощений E1-E38; и
b) полинуклеотида, кодирующего белки оболочки лентивируса.
E73. Способ по воплощению E72, дополнительно включающий экспрессирование последовательности полинуклеотида c), содержащего экзогенную нуклеиновую кислоту.
E74. Способ по воплощению E72 или E73, при этом полинуклеотид, содержащий конструкцию из нуклеиновой кислоты, дополнительно содержит последовательность нуклеиновой кислоты, кодирующей капсидные белки лентивируса.
E75. Способ по любому из воплощений E72-E74, дополнительно включающий извлечение лентивирусных частиц из клеток хозяина.
E76. Способ по любому из воплощений E72-E75, дополнительно включающий очистку лентивирусных частиц.
E77. Способ встраивания последовательности экзогенной нуклеиновой кислоты в геномную ДНК организма, включающий введение в организм лентивирусных частиц, содержащих конструкцию из нуклеиновой кислоты по любому из воплощений E1-E38 или слитый белок по любому из воплощений E40-E71 с тем, чтобы первый и второй ДНК-связывающие белки связались с определенной последовательностью геномной ДНК и вставили экзогенную нуклеиновую кислоту в геномную ДНК; причем экзогенная нуклеиновая кислота встраивается в определенную последовательность геномной ДНК.
E78. Способ контролируемого сайт-специфичного встраивания одной или нескольких копий последовательности экзогенной нуклеиновой кислоты в геном клеток, который включает:
a) введение в клетки слитого белка по любому из воплощений E40-E71 и
b) введение в клетки экзогенной нуклеиновой кислоты;
при этом связывание слитого белка с определенной последовательностью геномной ДНК в геноме клеток приводит к расщеплению генома и встраиванию одной или нескольких копий экзогенной нуклеиновой кислоты в геном этих клеток; причем слитый белок вводится в клетки при помощи лентивирусных частиц.
E79. Конструкция из нуклеиновой кислоты, включающая:
a) последовательность первого полинуклеотида, содержащего нуклеиновую кислоту, кодирующую первый ДНК-связывающий белок, сконструированный для связывания с определенной последовательностью геномной ДНК в геноме; причем первый ДНК-связывающий белок представляет собой цинк-пальцевый белок или белок Cas9;
b) последовательность второго полинуклеотида, содержащего нуклеиновую кислоту, кодирующую второй ДНК-связывающий белок, который обеспечивает встраивание экзогенной нуклеиновой кислоты в геном, причем второй ДНК-связывающий белок представляет собой:
(i) гиперактивную транспозазу PiggyBac или модифицированную гиперактивную PiggyBac с улучшенной специфичностью встраивания экзогенной нуклеиновой кислоты в геном по сравнению с гиперактивной PiggyBac либо
(ii) интегразу вируса иммунодефицита человека (ВИЧ) или модифицированную интегразу ВИЧ с улучшенной специфичностью встраивания экзогенной нуклеиновой кислоты в геном по сравнению с интегразой ВИЧ; и
c) необязательно последовательность полинуклеотида, содержащего нуклеиновую кислоту, кодирующую линкер;
причем конструкция из нуклеиновой кислоты кодирует слитый белок, включающий первый ДНК-связывающий белок, второй ДНК-связывающий белок и необязательно линкер между первым ДНК-связывающим белком и вторым ДНК-связывающим белком; а слитый белок обеспечивает встраивание экзогенной нуклеиновой кислоты в определенный сайт генома.
E80. Конструкция из нуклеиновой кислоты по воплощению E79, при этом белок Cas9 выбран из группы, состоящей из Cas9 человека, никазы Cas9 и «мертвого» Cas9.
E81. Конструкция из нуклеиновой кислоты по воплощению E79, при этом цинк-пальцевый белок представляет собой цинк-пальцевый белок типа C2H2, содержащий 6 доменов.
E82. Конструкция из нуклеиновой кислоты по любому из воплощений E79-E81, при этом линкер включает последовательность XTEN или последовательность GGS.
E83. Конструкция из нуклеиновой кислоты по любому из воплощений E79-E82, при этом 3′-конец последовательности первого полинуклеотида соединяется с 5′-концом второго полинуклеотида.
E84. Конструкция из нуклеиновой кислоты по любому из воплощений E79-E83, при этом: (a) первый ДНК-связывающий белок представляет собой белок Cas9 или цинк-пальцевый белок, а (b) второй ДНК-связывающий белок представляет собой гиперактивную транспозазу PiggyBac или модифицированную гиперактивную PiggyBac с улучшенной специфичностью встраивания экзогенной нуклеиновой кислоты в геном по сравнению с гиперактивной PiggyBac; причем конструкция из нуклеиновой кислоты содержит (c) последовательность полинуклеотида, содержащего нуклеиновую кислоту, кодирующую линкер, включающий последовательность XTEN или последовательность GGS, а 3′-конец последовательности первого полинуклеотида соединяется с 5′-концом второго полинуклеотида.
E85. Конструкция из нуклеиновой кислоты по любому из воплощений E79-E83, при этом: (a) первый ДНК-связывающий белок представляет собой белок Cas9 или цинк-пальцевый белок, а (b) второй ДНК-связывающий белок представляет собой интегразу ВИЧ или модифицированную интегразу ВИЧ с улучшенной специфичностью встраивания экзогенной нуклеиновой кислоты в геном по сравнению с интегразой ВИЧ; причем конструкция из нуклеиновой кислоты содержит (c) последовательность полинуклеотида, содержащего нуклеиновую кислоту, кодирующую линкер, включающий последовательность XTEN или последовательность GGS, а 3′-конец последовательности первого полинуклеотида соединяется с 5′-концом второго полинуклеотида.
E86. Конструкция из нуклеиновой кислоты по любому из воплощений E79-E84, при этом модифицированная гиперактивная транспозаза PiggyBac содержит мутации по одной или нескольким из числа аминокислот 245, 268, 275, 277, 287, 290, 315, 325, 341, 346, 347, 350, 351, 356, 357, 372, 375, 388, 409, 412, 432, 447, 450, 460, 461, 465, 517, 560, 564, 571, 573, 576, 586, 587, 589, 592 и 594 в соответствии с аминокислотной последовательностью гиперактивной PiggyBac по SEQ ID NO: 9.
E87. Конструкция из нуклеиновой кислоты по воплощению E86, при этом мутации модифицированной гиперактивной транспозазы PiggyBac включают одну или несколько модификаций аминокислот из числа R245A, D268N, R275A/R277A, K287A, K290A, K287A/K290A, R315A, G325A, R341A, D346N, N347A, N347S, T350A, S351E, S351P, S351A, K356E, N357A, R372A, K375A, R372A/K375A, R388A, K409A, K412A, K409A/K412A, K432A, D447A, D447N, D450N, R460A, K461A, R460A/K461A, W465A, S517A, T560A, S564P, S571N, S573A, K576A, H586A, I587A, M589V, S592G или F594L в соответствии с аминокислотной последовательностью гиперактивной PiggyBac по SEQ ID NO: 9.
E88. Конструкция из нуклеиновой кислоты по любому из воплощений E79-E84, при этом модифицированная гиперактивная транспозаза PiggyBac содержит мутации по одной или нескольким из числа аминокислот 245, 275, 277, 325, 347, 351, 372, 375, 388, 450, 465, 560, 564, 573, 589, 592, 594 в соответствии с аминокислотной последовательностью гиперактивной PiggyBac по SEQ ID NO: 9.
E89. Конструкция из нуклеиновой кислоты по воплощению E88, при этом мутации модифицированной гиперактивной транспозазы PiggyBac включают одну или несколько модификаций аминокислот из числа R245A, R275A, R277A, R275A/R277A, G325A, N347A, N347S, S351E, S351P, S351A, R372A, K375A, R388A, D450N, W465A, T560A, S564P, S573A, M589V, S592G или F594L в соответствии с аминокислотной последовательностью гиперактивной PiggyBac по SEQ ID NO: 9.
E90. Конструкция из нуклеиновой кислоты по воплощению E88, при этом модифицированная гиперактивная транспозаза PiggyBac имеет аминокислотную последовательность по SEQ ID NO: 9, где в положении 245 находится аминокислота A, в положении 275 – аминокислота R или A, в положении 277 – аминокислота R или A, в положении 325 – аминокислота A или G, в положении 347 – аминокислота N или A, в положении 351 – аминокислота E, P или A, в положении 372 – аминокислота R, в положении 375 – аминокислота A, в положении 450 – аминокислота D или N, в положении 465 – аминокислота W или A, в положении 560 – аминокислота T или A, в положении 564 – аминокислота P или S, в положении 573 – аминокислота S или A, в положении 592 – аминокислота G или S и в положении 594 – аминокислота L или F.
E91. Конструкция из нуклеиновой кислоты по воплощению E88, при этом модифицированная гиперактивная транспозаза PiggyBac имеет аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 120, 121, 122, 123, 124, 125, 126, 127, 128 и 129.
E92. Конструкция из нуклеиновой кислоты по воплощению E88, при этом модифицированная гиперактивная транспозаза PiggyBac имеет аминокислотную последовательность, которая по меньшей мере на 80% идентична последовательности, выбранной из группы, состоящей из SEQ ID NO: 119, 120, 121, 122, 123, 124, 125, 126, 127, 128 и 129, причем модифицированная гиперактивная PiggyBac проявляет большую специфичность встраивания ДНК в геном по сравнению с гиперактивной PiggyBac.
E93. Конструкция из нуклеиновой кислоты по любому из воплощений E79-E83 или E85, при этом модифицированная интеграза ВИЧ содержит мутации по одной или нескольким из числа аминокислот 10, 13, 64, 94, 116, 117, 119, 120, 122, 124, 128, 152, 168, 170, 185, 231, 264, 266 или 273 в соответствии с аминокислотной последовательностью интегразы ВИЧ дикого типа по SEQ ID NO: 1.
E94. Конструкция из нуклеиновой кислоты по воплощению E93, при этом мутации модифицированной интегразы ВИЧ включают одну или несколько из мутаций D10K, E13K, D64A, D64E, G94D, G94E, G94R, G94K, D116A, D116E, N117D, N117E, N117R, N117K, S119A, S119P, S119T, S119G, S119D, S119E, S119R, S119K, N120D, N120E, N120R, N120K, T122K, T122I, T122V, T122A, T122R, A124D, A124E, A124R, A124K, A128T, E152A, E152D, Q168L, Q168A, E170G, F185K, R231G, R231K, R231D, R231E, R231S, K264R, K266R или K273R в соответствии с аминокислотной последовательностью интегразы ВИЧ дикого типа по SEQ ID NO: 1.
E95. Вектор, содержащий конструкцию из нуклеиновой кислоты по любому из воплощений E79-E95, при этом вектор подходит для экспрессии в клетках млекопитающих, дрожжевых клетках, клетках насекомых, клетках растений, грибковых клетках или клетках водорослей.
E96. Клетка-хозяин, содержащая конструкцию из нуклеиновой кислоты или вектор по любому из воплощений E79-E95.
E97. Слитый белок, полученный при экспрессии конструкции из нуклеиновой кислоты по любому из воплощений E79-E94.
E98. Композиция, включающая конструкцию из нуклеиновой кислоты, вектор или слитый белок по любому из воплощений E79-E95 или E97 и последовательность полинуклеотида, кодирующего экзогенную нуклеиновую кислоту для вставки в геном, причем композиция содержится в упаковочном векторе или связана с ним.
E99. Композиция по воплощению E98, при этом конструкция из нуклеиновой кислоты находится в виде РНК, ДНК или белка, а последовательность полинуклеотида, кодирующего экзогенную нуклеиновую кислоту, находится в виде ДНК или РНК.
E100. Композиция по любому из воплощений E98-E99, при этом упаковочный вектор представляет собой наночастицы или лентивирусные частицы.
E101. Способ контролируемого сайт-специфичного встраивания одной или нескольких копий последовательности экзогенной нуклеиновой кислоты в клетки, который включает: (a) введение в клетки конструкции из нуклеиновой кислоты, вектора или слитого белка по любому из воплощений E79-E95 или E97 и (b) введение в клетки экзогенной нуклеиновой кислоты; при этом связывание слитого белка с определенной последовательностью геномной ДНК в геноме клеток приводит к расщеплению генома и встраиванию одной или нескольких копий экзогенной нуклеиновой кислоты в геном этих клеток.
E102. Модифицированная гиперактивная транспозаза PiggyBac, имеющая аминокислотную последовательность по SEQ ID NO: 9, где в положении 245 находится аминокислота A, в положении 275 – аминокислота R или A, в положении 277 – аминокислота R или A, в положении 325 – аминокислота A или G, в положении 347 – аминокислота N или A, в положении 351 – аминокислота E, P или A, в положении 372 – аминокислота R, в положении 375 – аминокислота A, в положении 450 – аминокислота D или N, в положении 465 – аминокислота W или A, в положении 560 – аминокислота T или A, в положении 564 – аминокислота P или S, в положении 573 – аминокислота S или A, в положении 592 – аминокислота G или S и в положении 594 – аминокислота L или F.
E103. Модифицированная гиперактивная транспозаза PiggyBac по воплощению E102, которая имеет аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 120, 121, 122, 123, 124, 125, 126, 127, 128 и 129.
E104. Модифицированная гиперактивная транспозаза PiggyBac по воплощению E102, имеющая аминокислотную последовательность, которая по меньшей мере на 80% идентична последовательности, выбранной из группы, состоящей из SEQ ID NO: 119, 120, 121, 122, 123, 124, 125, 126, 127, 128 и 129, причем модифицированная гиперактивная PiggyBac проявляет большую специфичность встраивания ДНК в геном по сравнению с гиперактивной PiggyBac.
Содержание всех цитируемых ссылок (включая ссылки на литературу, патенты, патентные заявки и веб-сайты), которые могут цитироваться в этой заявке, прямо включено сюда путем ссылки во всей полноте для любых целей так же, как и ссылки, приведенные в них. Следующие примеры представлены в качестве иллюстрации, а не для ограничения.
Примеры
“PB” и “hyPB” применяются взаимозаменяемым образом для обозначения гиперактивной транспозазы PiggyBac. Приведенные ниже примеры 1-3 касаются получения и эффективности с точки зрения направленного встраивания конструкций слитых белков программируемых транспозаз и Cas9. В примере 1 были успешно получены различные ДНК конструкции для транспозаз гиперактивной PiggyBac и Sleeping Beauty, слитые с различными вариантами Cas9, вызывающие встраивание транспозона в геном трансфицированных клеток. Примечательно, что конструкции для PiggyBac и Cas9 были способы обеспечить направленное встраивание в представляющий интерес сайт генома (Пример 2). В примере 3 представлены модифицированные транспозазы, созданные для повышения специфичности встраивания последовательности экзогенной нуклеиновой кислоты в геном.
Пример 1. ДНК-векторы для экспрессии слитых белков программируемых транспозаз
Целью данного эксперимента была проверка различных конфигураций слияния гиперактивных транспозаз PiggyBac (обозначены здесь как hyPB или PB) и Sleeping Beauty (обозначены здесь как SB100x) с нуклеазой (h), никазой (n) и «мертвым» (d) Cas9 на эффективность встраивания транспозонов. Получали слитые белки программируемых транспозаз путем включения в экспрессирующий вектор pcDNA3.3-TOPO (остов плазмиды Invitrogen, плазмида Addgene #41815) последовательностей ДНК, кодирующих Cas9 дикого типа человека (hCas9), никазу Cas9 (nCas9) или «мертвый» Cas9 (dCas9) (SEQ ID NO: 64-66, соответственно) и гиперактивную транспозазу PiggyBac (PB) или Sleeping Beauty (SB100) (SEQ ID NO: 67-68, соответственно). Создавали векторы, в которых 3′-конец Cas9 соединяется с 5′-концом каждой из транспозаз линкерной последовательностью нуклеиновой кислоты (SEQ ID NO: 48), кодирующей линкер GGS (hCas9PB, nCas9PB, dCas9PB, hCas9SB, nCas9SB и dCas9SB). Создавали и другие векторы, в которых 3′-конец каждой из транспозаз соединяется с 5′-концом Cas9 линкерной последовательностью нуклеиновой кислоты (SEQ ID NO: 48), кодирующей линкер GGS (PBhCas9, PBnCas9, PBdCas9, SBhCas9, SBnCas9 и SBdCas9). В таблице 2 представлена сводка по слитым конструкциям.
Таблица 2. Список белков программируемых транспозаз, полученных в примере 1
Перед трансфекцией быстро оттаивали замороженные клетки HEK293T при 37°C, а затем ресуспендировали в 5 мл предварительно нагретой среды и осаждали центрифугированием при 1000 об/мин в течение 4 мин. Осадок ресуспендировали в свежей среде и вносили ~1,6×106 клеток в новую колбу T75. Когда клетки достигали конфлюэнтности 95%, их пассировали с помощью трипсина и высеивали при конфлюэнтности 40%. Клетки пассировали дважды перед использованием в экспериментах.
Для экспериментов по трансфекции высеивали по 5×105 клеток HEK293T на лунку в многолуночный планшет с полной средой DMEM (модифицированная Дюльбекко среда Игла (DMEM) с добавлением 10% фетальной телячьей сыворотки, 2 мМ глутамина и 100 ед./мл пенициллина/0,1 мг/мл стрептомицина). Перед трансфекцией среду заменяли на 2,7 мл свежей полной среды DMEM. Смешивали среду Opti-MEM I с пониженным содержанием сыворотки с каждой комбинацией плазмид и с раствором линейного полиэтиленимина (PEI 25K) 1 мг/мл. Использовали соотношение PEI 25K (мкг):общая ДНК (мкг) = 3:1. Оба раствора смешивали и инкубировали при комнатной температуре в течение 15 мин. После инкубации в клетки по каплям вносили 300 мкл смеси. Через 24 ч после трансфекции среду заменяли на свежую полную среду. Клетки после трансфекции собирали для проточной цитометрии или сортировки клеток и экстракции ДНК.
Клетки HEK293T трансфицировали совместно плазмидой, кодирующей слитый белок программируемой транспозазы из таблицы 2, плазмидой, кодирующей нуклеиновую кислоту для встраивания, а именно транспозон с RFP (красный флуоресцентный белок) или GFP (зеленый флуоресцентный белок), и направляющей РНК, нацеленной на сайт AAVS1 (сайт-1 встраивания аденоассоциированного вируса) в геноме человека. В качестве положительного контроля использовали гиперактивные PiggyBac и SB100, а транспозон сам по себе – в качестве отрицательного контроля для выявления эписомальной экспрессии (т.е. экспрессии из невстроившихся плазмид). Анализировали флуоресценцию методом проточной цитометрии вплоть до 14-го дня, после чего эписомальная флуоресценция не обнаруживалась. Затем клетки подвергали сортингу по экспрессии GFP, а через два дня после сортинга определяли встраивание целевой ДНК путем подсчета процента флуоресцентных клеток.
Результаты и выводы. На фиг. 1A и фиг. 1C представлены результаты по слитому Cas9-PB, а на фиг. 1B – результаты по слитому Cas9-SB100. Cas9 человека, слитый с гиперактивной PiggyBac (hCas9PB), и никаза Cas9, слитая с гиперактивной PiggyBac (nCas9PB), повышали процент флуоресцентных клеток примерно на 8% по сравнению с отрицательным контролем – эписомальным RFP через 14 дней (фиг. 1A, 1C). Следовательно, данные слитые белки способны успешно встраивать экзогенную ДНК в геном клетки. А исследованные слитые белки Cas9-Sleeping Beauty давали не больше флуоресцентных клеток, чем отрицательный контроль – эписомальный GFP через 14 дней (фиг. 1B).
Пример 2. Эффективность направленной транспозиции слитых белков программируемых транспозаз
Вслед за предыдущим примером исследовали, будет ли вставка направленной (или не направленной) при тех конфигурациях, которые в целом давали самые лучшие вставки в Примере 1. Для этого клетки HEK293T трансфицировали совместно с помощью липофектамина 3000 плазмидой (pSico), кодирующей hCas9PB или nCas9PB, плазмидой Genetrap, кодирующей транспозон с инвертированными повторами и GFP без промотора, и направляющей РНК, нацеленной на сайт AAVS1 или сайт в гене CD46 после промотора в геноме человека. 3′-Конец Cas9 соединяли с 5′-концом транспозазы при помощи линкера (SEQ ID NO: 48). Пример структуры экспрессирующего Cas9PB вектора представлен на фиг. 2A. Транспозаза содержала акцептор сплайсинга и GFP без промотора между 3′- и 5′-повторами. направляющая РНК и Cas9 направляют транспозазу для встраивания транспозона в район промотора. При таком подходе клетки становятся флуоресцентными, только если транспозон будет вставлен в целевой сайт.
Результаты и выводы. Определение процента экспрессирующих GFP клеток показало, что слитые белки с программируемой транспозазой Cas9-PiggyBac (“прицельный hCas9”) и никазой Cas9-PiggyBac (“прицельный nCas9”) дают лучшую направленную доставку целевой ДНК по сравнению с контролями “неприцельный” (контроль на вставку вообще (только PiggyBac)) и “эписомальный” (отрицательный контроль без встраивания (только транспозон)) (фиг. 2B). При этом повышение сигнала в 3 раза и 4 раза выше фона было значимым, особенно с учетом того, что не все клетки были эффективно трансформированы всеми векторами, необходимыми для встраивания транспозона; а эффективность случайной вставки hyPB в неоптимизированных условиях, которые использовались здесь, составляет 10-15%.
Пример 3. Получение модифицированных гиперактивных транспозаз PiggyBac
Создавали модифицированные гиперактивные транспозазы PiggyBac для повышения специфичности встраивания последовательности экзогенной нуклеиновой кислоты в геном. Список мутаций аминокислот транспозазы приведен в таблице 3.
Таблица 3. Сайты мутаций для гиперактивной PiggyBac относительно последовательности гиперактивной PiggyBac SEQ ID NO: 9
Далее в примере 4 создавали несколько конструкций с тем, чтобы цинк-пальцевый белок (ZFP) мог связываться с хромосомным сайтом мишени для вставки представляющего интерес гена. ZFP составляет альтернативу Cas9 в качестве ДНК-связывающего белка. Примеры 5-13 в общем касаются получения и эффективности с точки зрения направленного встраивания конструкций слитых белков интегразы ВИЧ-1 и Cas9/ZFP. В частности, в примере 5 получали слитые белки ZFP и интегразы. В примерах 6-10 представлены различные упаковочные системы, дефектные по интегразе (то есть это невстраивающие векторы), призванные служить в качестве основы для исследований in vitro, свидетельствующих о восстановление функции встраивания со слитыми белками интегразы, созданными в примере 11. В примере 12 отмечается, что направленные слитые белки интегразы повышают процент направленного встраивания.
Пример 4. Получение направленного цинк-пальцевого белка (ZFP)
Целью было создание таких ZFP, которые связываются с сайтом мишенью на хромосоме для вставки представляющего интерес гена. Был получен 6-доменный цинк-пальцевый белок для посадки на сайт AAVS1 (SEQ ID NO: 40) в геноме человека. В таблице 4 представлены последовательности ДНК мишени и соответствующие спирали ZFP. Были получены конструкции, кодирующие сайты мишени и ZFP (AAVS1-6d-ZFP). Последовательности нуклеиновой кислоты, кодирующие ZFP, и аминокислотные последовательности ZFP представлены в SEQ ID NO: 32 и 33, соответственно.
Таблица 4. Список сайтов мишени AAVS1 и соответствующих спиралей ZFP
Пример 5. Получение слитых белков ZFP-интеграза
Получали слитые белки интегразы с ZFP, содержащими 6 доменов (эффективно специфичные для последовательности). Для получения сайт-специфичной интегразы клонировали ZFP, полученный в примере 4 (AAVS1-6d-ZFP), в экспрессирующий вектор pcDNA3.1 вместе с интегразой ВИЧ-1 (SEQ ID NO: 1) (pZFP-AAVS1-6d-IN). Последовательность, кодирующая слитый белок, содержит N-концевой сигнал ядерной локализации (SEQ ID NO: 47) и последовательность линкера GGS (SEQ ID NO: 48) между ZFP и интегразой (фиг. 3).
Получали и другие векторы для слияния интегразы, как-то pZFP-TRCa-IN (включающий SEQ ID NO: 38, нацеленную на локус TRCa) и pZFP-AAVs1-TEX-IN (включающий линкер TEX (SEQ ID NO: 61)), которые получали такими же методами.
Пример 6. Получение дефектных по интегразе ДНК-векторов
Создавали дефектные по интегразе упаковочные системы, служащие в качестве основы для исследований in vitro с использованием сконструированной интегразы. Дефектные по интегразе конструкции получали из невстраивающейся упаковочной плазмиды (NILV) psPAX2. Плазмиды psPAX2 содержат одиночную мутацию N64D и двойные мутации N64D/N116D. Создавали и плазмиды с делецией интегразы (ΔIN), лишенные всей кодирующей области интегразы. Создавали некодирующие плазмиды, содержащие стоп-кодон перед кодирующей последовательностью интегразы (пример 8 ниже). Создавали и плазмиды, содержащие укороченные интегразы, включая конструкцию, содержащую С-концевой домен и ДНК-связывающий домен без cPPT/CTS (пример 10 ниже). Следовали общим методикам клонирования, как описано вкратце ниже.
Методика KAPA HiFi HotStart
Для экспериментов по ПЦР с использованием KAPA HiFi HotStart готовили реакционную смесь для ПЦР по методике производителя набора KAPA HiFi PCR Kit. Реакции ПЦР KAPA HiFi проводили на установке MasterCycler Pro.
Выделение плазмидной ДНК
Плазмидную ДНК выделяли с помощью набора QIAprep Spin Miniprep Kit по методике производителя. Собирали бактериальные культуры центрифугированием при 5000 об/мин в течение 3 мин. Осадки клеток ресуспендировали в 250 мкл буфера P1 и смешивали с 250 мкл буфера P2, переворачивая пробирки 4-6 раз. Добавляли 350 мкл буфера N3 и перемешивали, переворачивая пробирки. Пробирки Eppendorf центрифугировали 10 мин при 12000 об/мин для удаления обломков клеток и хромосомной ДНК. Супернатанты переносили в прилагаемые спин-колонки QIAprep и центрифугировали в течение 1 мин (12000 об/мин). Образцы дважды промывали 0,5 мл буфера PB и 0,75 мл буфера PE и каждый раз центрифугировали по 1 мин при 12000 об/мин. Дополнительным центрифугированием в течение 1 мин при 12000 об/мин удаляли остатки буфера из промывочного раствора. Спин-колонки QIAprep переносили в новые микроцентрифужные пробирки на 1,5 мл и добавляли 50 мкл воды для элюирования плазмиды, оставляя пробирки на 1 мин и затем центрифугируя в течение 1 мин при 12000 об/мин. Измеряли концентрации с помощью NanoDrop One.
Выделение и очистка плазмидной ДНК
Штаммы бактерий (DH5α или DH10B), содержащие требуемые плазмиды, культивировали в течение ночи в среде LB, содержащей 100 мкг/мл карбенициллина. Выделяли плазмиды с помощью наборов Mini или Maxi фирмы NZYTech по методике производителя. Плазмиды элюировали в 30 мкл (miniprep) либо 500 мкл (maxiprep) горячей воды при 65°C. Плазмиды хранили при -20°C. Для очистки на ПЦР обрабатывали реакционную смесь с помощью набора для очистки на ПЦР. Элюировали ДНК в 30 мкл горячей воды при 65°C.
Гель-электрофорез ДНК
Растворяли агарозу в 100 мл буфера TAE путем кипячения. В жидкие гели добавляли 4 мкл Greensafe на 100 мл раствора агарозы и заливали в лоток. Для визуализации препаратов ДНК смешивали ДНК с 6-кратным красителем и наносили на 1% агарозный гель. Кроме того, в одну камеру вносили по 1 мкл маркера gene ladder на 1 мм дорожки геля. Гели разгоняли в течение 1,5 ч при 100 В и визуализировали с помощью трансиллюминатора.
Трансформация
Для экспериментов по трансформации с клетками DH5α трансформировали 50 мкл клеток DH5α плазмидами по методике производителя. После восстановления в среде s.o.c. бактерии осаждали при 15000×g в течение 30 сек и ресуспендировали в 50 мкл среды LB. Клетки наносили шпателем на чашки с LB-агаром, содержащим 100 мкг/мл карбенициллина, и инкубировали при 37°C в течение ночи. Отбирали колонии и инокулировали на всю ночь в среду LB, содержащую 100 мкг/мл карбенициллина. Жидкие культуры снова использовали либо для выделения плазмид, либо для хранения в глицерине. Для хранения в глицерине смешивали 500 мкл жидкой культуры с 500 мкл 50% глицерина и хранили при -80°C.
Для экспериментов по трансформации с ультракомпетентными клетками XL-10 Gold клетки сначала оттаивали на льду и вносили по 45 мкл клеток в охлажденные полипропиленовые круглодонные пробирки Falcon на 14 мл. К клеткам добавляли по 2 мкл смеси β-ME, поставляемой с набором. Содержимое пробирок осторожно взбалтывали и инкубировали клетки на льду в течение 10 мин (побалтывая через каждые 2 мин). В каждую порцию клеток добавляли 1,5 мкл обработанной DpnI ДНК, перемешивали и инкубировали на льду в течение 30 мин. Смеси клеток и ДНК в пробирках быстро нагревали до 42°C на 30 сек. Затем пробирки инкубировали на льду в течение 2 мин. В каждую пробирку добавляли 0,5 мл нагретого до 42°C бульона NZY+ и инкубировали 1 час при 37°C со встряхиванием при 225-250 об/мин. Затем смеси высеивали на чашки с агаром, содержащим соответствующие антибиотики для плазмидных векторов. Отбирали 5 колоний для выделения ДНК и проверяли последовательности. Отбирали и сохраняли колонию № 1.
Пример 7. Получение невстраивающихся векторов, содержащих PPT или ZFP-модифицированные слитые белки интегразы
Для получения дефектных по интегразе (IN), но в остальном полностью функциональных плазмид psPAX2 клонировали домен полипиримидинового тракта (PPT) (SEQ ID NO: 74), который важен для последующего образования двухцепочечной кДНК во всех РНК-геномах ретровирусов типа лентивирусов), в вектор psPAX2, не содержащий интегразы (psPAX2-ΔIN). В psPAX2-ΔIN клонировали синтетическую цинк-пальцевую конструкцию, нацеленную на AAVS1, полученную в примере 4 (AAVS1-6d-ZFP-IN). Были составлены два разных прямых праймера и один и тот же обратный праймер (SEQ ID NO: 75-77) для PPT со стоп-кодоном и без него (IN+PPT и IN+PPT(STOP)). Были составлены два разных прямых праймера и один и тот же обратный праймер (SEQ ID NO: 78-80) для AVS1-6d-ZFP-IN с сигналом ядерной локализации и без него (AAVS1-6d-ZFP-IN и AAVS1-6d-ZFP-IN(-NLS)). Вставки амплифицировали по ПЦР при стандартных условиях KAPA, температуре отжига 62°C и времени элонгации 40 сек для PPT и 90 сек для AAVS1-6d-ZFP-IN. Продукты ПЦР разделяли методом гель-электрофореза.
Амплифицированные продукты очищали и проводили сборку по методике при соотношении 1:2,5 остова:вставка и 5 циклах. Трансформировали 50 мкл компетентных клеток продуктами лигирования по 4 мкл и высеивали 60% компетентных клеток на чашки с карбенициллином. Первоначальную проверку колоний проводили методами рестрикционного расщепления и гель-электрофореза ДНК. Были отобраны следующие колонии: колонии 1 и 2 (IN+PPT F1+R, AAVS1-6d-ZFP-IN F1+R, AAVS1-6d-ZFP-IN(-NLS) F2 +R) и колонии 7 и 8 (IN+PPT(STOP) F2+R). Для дальнейшей проверки наличия в колониях правильной вставки проводили ПЦР колоний при 4 мМ Mg, 62-STS со стандартной Taq-полимеразой NEB.
Пример 8. Получение невстраивающихся векторов путем вставки стоп-кодона
Невстраивающиеся векторы получали путем вставки стоп-кодона перед открытой рамкой считывания интегразы (psPAX2-TAA-IN). psPAX2-TAA-IN получали посредством сайт-направленного мутагенеза путем добавления двух стоп-кодонов после сайта расщепления для протеазы в начале интегразы. Для получения psPAX2-TAA-IN использовали условия ПЦР для сайт-направленного мутагенеза.
После реакции ПЦР пробирки ставили на лед на 2 минуты для охлаждения. Затем прямо в каждую реакцию амплификации добавляли 1 мкл DpnI и инкубировали при 37°C в течение 5 мин для расщепления исходной (немутантной) двухцепочечной ДНК.
Для проверки того, что сайт-направленный мутагенез не давал каких-либо нежелательных модификаций, расщепляли плазмидную ДНК. Расщепление psPAX2 и psPAX2-TAA-IN с помощью SacI и AgeI должно давать три полосы в 7500, 1900 и 1300 п.н. Расщепление psPax2-ΔIN с помощью SacI и AgeI должно давать три полосы в 7500, 1300 и 800 п.н. Проводили реакции расщепления, в результате которых получили правильный профиль полос.
Пример 9. Внедрение интегразы дикого типа в дефектный по интегразе вектор
Целью была разработка методологии для проверки того, может ли невстраивающийся вектор восстановить свою встраивающую активность при экспрессии различных форм слитых белков интегразы. Для проверки того, что вектор psPAX2-ΔIN был полностью функциональным, в него вставляли интегразу методом сборки по Гибсону. Кроме того, для проверки того, что сайты сборки подходят для клонирования слитой “IN”, клонировали IN дикого типа с дополнительным N-концом IN, вставленным в остов перед этим сайтом (с Leu, которого там не должно быть). Это также делали с дополнительной последовательностью мишени для протеазы, чтобы избежать этого фальшивого N-концевого домена. Для амплификации фрагментов IN-1, IN-2 и IN-3 проводили реакции ПЦР.
Амплифицированные продукты ПЦР разделяли методом гель-электрофореза ДНК. Выделяли амплифицированные полосы и проводили сборку при соотношении 1:2,5 остова:вставка и 5 циклах при 37°C. Трансформировали 50 мкл компетентных клеток продуктами лигирования по 4 мкл и высеивали их на чашки с карбенициллином.
Для получения конструкции, содержащей IN-3, проводили сборку Гибсона по стандартной методике из набора для 1-стадийной сборки Gibson Assembly HiFi (с использованием CRG MM)(SGI-DNA, Inc., www.sgidna.com/products/gibson-assembly-reagents/). Готовили реакционные смеси и проводили сборку в течение 1 ч при 50°C. Компетентные клетки трансформировали 2 мкл реакционной смеси.
Трансформировали 50 мкл компетентных клеток продуктами лигирования по 2 мкл и высеивали их на чашки с карбенициллином.
Пример 10. Получение невстраивающихся векторов, содержащих интегразу с укороченным C-концевым доменом
В вектор psPAX2 клонировали С-концевой домен (CTD) (нуклеотиды 83-118 по SEQ ID NO: 74) и фрагменты интегразы CppT + CTD (SEQ ID NO: 74).
Амплифицированные продукты ПЦР разделяли методом гель-электрофореза ДНК. Проводили лигирование CppT+CTD в условиях, использовавшихся в примере 9.
Проводили лигирование по 5 циклов при 65°C и трансформацию продуктами лигирования. Колонии не выросли. Снова проводили лигирование и трансформацию, и три колонии проверяли путем секвенирования с праймером IN-fw (SEQ ID NO: 81).
Пример 11. Получение слитых белков интегразы
Создавали направленные слитые белки интегразы путем включения в экспрессирующий вектор pcDNA3.3 интегразы ВИЧ-1 и направленного ZFP либо Cas9 человека. Получили один вектор, в котором 3′-конец ZFP или Cas9 соединяется с 5′-концом интегразы линкером из нуклеиновой кислоты. Получали другой вектор, в котором 3′-конец интегразы соединяется с 5′-концом ZFP или Cas9 линкером из нуклеиновой кислоты. Использовали линкеры XTEN или GGS длиной в 13, 16, 19, 22, 25 или 28 аминокислот. Слитый белок ZFP-интеграза был сконструирован для посадки на сайт AAVS1 или в локус T-клеточного рецептора альфа (TCRα) в геноме человека. Слитый белок Cas9-интеграза использовали в комбинации с направляющими РНК, нацеленными на сайт AAVS1 или на локус TCRα в геноме человека. В таблице 5 представлен список слитых белков модифицированной интегразы.
Таблица 5. Список слитых белков модифицированной интегразы, полученных в примере 11
Пример 12. Цис- и транс-комплементация дефектного по интегразе лентивируса с помощью направленных слитых белков интегразы
Направленные слитые белки интегразы из Примера 11 использовали для комплементации отсутствия способности к встраиванию у невстраивающегося лентивируса, экспрессирующего IN с двумя мутациями в каталитическом домене (D64V/D116N). Для этого эксперимента клонировали направленные слитые белки интегразы в вектор pcDNA3.1. Лентивирус получали при совместной трансфекции клеток с помощью pSICO (со вставкой для экспрессии GFP), pMD2.G (VSVG для экспрессии оболочки), PAX2 (содержащего упаковывающие белки и интегразу) или NILV-PAX2 (содержащего упаковывающие белки) и вектора pcDNA3.1, содержащего либо интегразу дикого типа, либо направленные интегразы (таблица 6).
Таблица 6. Условия для комплементации дефектного по интегразе лентивируса с помощью направленных слитых белков интегразы
плазмида
IN(AAVS1)
IN(AAVS1)
Высеивали по 6×105 клеток HEK293T (8-й пассаж) на лунку в 6-луночный планшет и инкубировали в течение ночи. За 5 часов до начала продукции вируса среду заменяли на 1,7 мл среды, содержащей 1:1000 хлорохина дифосфата (CD; исходный раствор = 25 мМ). Плазмиды трансфицировали при молярном соотношении 1,6:1,32:0,72:3,32 (pSICO: PAX2:VSVG: wtIN-rescue). В качестве реагента для трансфекции использовали PEI (полиэтиленимин; исходный раствор = 1 мг/мл), по 3 мкл PEI на 1 мкг общей ДНК, используемой для трансфекции. ДНК разводили в 83 мкл Opti-MEM и 83 мкл PEI, перемешивали и инкубировали в течение 15-20 мин при комнатной температуре. Каждую смесь для трансфекции добавляли по каплям к клеткам со средой CD. Клетки инкубировали в течение ночи, а на следующий день среду заменяли на 2,5 мл свежей среды. На следующий день супернатанты клеток центрифугировали 5 мин при 1000 об/мин и пропускали через фильтр на 45 мкм. Супернатанты, содержащие вирус, хранили при -80°C.
Первым шагом была проверка того, что различные упаковки лентивируса сохраняют способность инфицировать клетки независимо от их содержимого. Для определения титра вируса высеивали по 75 000 клеток HEK293T на лунку в 6-луночный планшет. Клетки инфицировали смесью из 1 мл среды, содержащей полибрен 1:100, и 500 мкл полученного ранее супернатанта вируса (1:3). Среду меняли на следующий день. А на следующий день среду отсасывали и отделяли клетки с помощью 200 мкл трипсина. Реакцию останавливали добавлением 800 мкл нормальной среды и анализировали методом проточной цитометрии. Определяли титры вируса для лентивируса интегразы дикого типа (LV), пустых вирусных частиц (LVO), невстраивающегося лентивируса (NILV), невстраивающегося лентивируса с интегразой дикого типа (NILV+IN), невстраивающегося лентивируса со слитым белком ZFP-интеграза (NILV+ZP-IN(AAVS1)), невстраивающегося лентивируса со слитым белком Cas9-интеграза (NILV+Cas-IN) и лентивируса интегразы дикого типа с интегразой дикого типа (LV+IN). В качестве положительного и отрицательного контроля использовали LV и LVO, соответственно. Инфицировали клетки HEK293T и определяли титр вируса путем подсчета числа GFP-положительных клеток (фиг. 4). Результаты. Титр вируса был в пределах одного порядка величины для всех условий.
Затем определяли общую способность к встраиванию у направленных слитых белков интегразы методами проточной цитометрии и секвенирования следующего поколения для целевой вставки. Клетки HEK293T инфицировали при одной и той же множественности заражения для всех условий и измеряли флуоресценцию GFP через 3, 5, 7, 10 и 12 дней после инфицирования. Через 7 дней после инфицирования клетки подвергали сортингу по экспрессии GFP. Результаты. На 12-й день клетки, инфицированные NILV без комплементации, имели меньший процент клеток, экспрессирующих GFP (фиг. 5), что означает снижение способности к продукции вируса.
Для оценки способности к направленному встраиванию у исследуемых слитых белков интегразы выделяли геномную ДНК по методике из набора DNeasy Blood and Tissue Kit (Qiagen) на 12-й день. Культуры клеток собирали центрифугированием при 190 об/мин в течение 5 мин (максимум 5×105). Осадки суспендировали в 200мкл PBS (фосфатно-солевого буфера). Добавляли 20 мкл протеиназы K вместе с 200 мкл буфера AL. После вибромешалки образцы инкубировали при 56°C в течение 10 мин. После добавления 200 мкл этанола (96-100%) и краткого встряхивания смеси переносили в спин-колонки DNeasy Mini, помещали в пробирки для сбора на 3 мл и центрифугировали при 8000 об/мин в течение 1 мин. Спин-колонки переносили в новые пробирка для сбора на 2 мл и добавляли 500 мкл буфера AW1. Пробирки центрифугировали при 8000 об/мин в течение 1 мин. Эту операцию промывки повторяли для буфера AW2 (центрифугирование 3 мин). Затем спин-колонки переносили в новые микроцентрифужные пробирки на 1,5 мл и добавляли 200 мкл буфера AE в центр мембраны спин-колонки для элюирования ДНК, пробирки выдерживали 1 мин, после чего центрифугировали 1 мин при 8000 об/мин. Концентрацию геномной ДНК определяли с помощью NanoDrop One.
Проводили обратное клонирование с олигонуклеотидами, специфичными для вставленных вирусных LTR. Проводили целевое секвенирование следующего поколения и анализировали по следующим параметрам: фильтровать прочтения так, чтобы R1 и R2 содержали соответствующий праймер для секвенирования, ограничить проверку крайними левыми нуклеотидами (столько же п.н., как у праймера), допускается 2 несовпадения, обрезать последовательности праймеров (SEQ ID NO: 82-89), фильтровать прочтения так, чтобы R1 и R2 содержали соответствующие основания LTR, ограничить проверку 5 крайними левыми нуклеотидами прочтения, использовать 5 первых оснований LTR (после праймера для секвенирования) с K = 3 (это значит, что для последовательности ACTGA следует проверять наличие в прочтении одного из следующих k-меров: ACT, CTG, TGA), допуская 2 несовпадения, обрезать соответствующие пары нуклеотидов LTR, картировать прочтения по эталонному геному, получить охват (число прочтений на 1 сайт вставки), делить на 2 те участки, где перекрываются R1 и R2, добавить только один из сайтов вставки, если нет перекрывания R1 и R2, применить порог охвата, рассчитать охват на каждые 10 млн. нуклеотидов эталонного генома и построить графики охвата, рассчитать процент охвата для каждого сайта вставки. Результаты. Направленные слитые белки интегразы повышают охват сайта AAVS1 и процент вставок в цель (таблица 7 и фиг. 6). Как видно из таблицы 7, когда вставка осуществляется слитыми белками интегразы, количество прочтений на целевом сайте больше по сравнению с IN дикого типа, что указывает на направленность вставки. На фиг. 6 представлены наиболее частые целевые сайты в геноме для IN и ZFP_IN (AAVS1), что означает наличие направленной вставки только в слитом состоянии.
Таблица 7. Количество прочтений по AAVS1 и процент вставок в цель у направленных слитых белков интегразы
по AAVS1
Также создавали и второй ZFP для посадки на сегмент нуклеиновой кислоты гена CCR5. Этот цинк-пальцевый белок сливали с интегразой ВИЧ-1, получая интегразу, нацеленную на CCR5. Лентивирус, содержащий эту ZFP-IN, получали, как описано выше, и трансдуцировали им клетки HEK293T (NILV+ZP-IN(CCR5) (таблица 6). Результаты. Титр вируса NILV+ZP-IN(CCR5) был близок к LV и NILV+IN (фиг. 7A). Эта конструкция вырабатывала вирусные частицы с такой же эффективностью, что и другой исследованный слитый белок ZFP_IN (фиг. 7B и C). Его способность к встраиванию в ДНК сайт-специфичным образом не проверяли на CCR5.
В другом эксперименте недавно клонированные экспрессирующие векторы для слитой ZFP-IN с 6d были нацелены на локус TCRα и направляющая РНК нацелена на тот же сайт (см. Пример 11). При анализе проверяли, сможет ли интеграза дикого типа и слитая с ZFP-интеграза комплементировать способность к встраиванию у NILV и способствовать избирательному встраиванию кассеты CAR-T. Клетки Jurkat инфицировали при одной и той же множественности заражения для всех частиц, нацеленных на вставку в TCRα. В этом эксперименте вирусные частицы нагружали кассетой CAR-T CD19, что должно привести к потере экспрессии белка CD3 (кодируемого геном TCRα) после направленной вставки. Отслеживали процент CD19-положительных и CD3-отрицательных клеток по времени. Титр лентивируса представлен на фиг. 8A, а процент клеток, экспрессирующих CAR на 3-й и 14-й день, представлен на фиг. 8B. Процент экспрессирующих CD3 клеток представлен на фиг. 8C. Это означает, что транскомплементация не работает в контексте этой линии клеток в отсутствие VPR, важного фактора для эффективной транскомплементации IN.
Пример 13. Получение модифицированных интеграз при помощи сайт-направленного мутагенеза и насыщающего мутагенеза
Получали модифицированные интегразы при помощи сайт-направленного мутагенеза и насыщающего мутагенеза. При сайт-направленном мутагенезе модифицированные интегразы ВИЧ-1 получали путем мутации аминокислот при помощи сайт-направленного мутагенеза. Использовали набор для направленного мутагенеза QuikChange Lightning Multi Site-Directed Mutagenesis Kit и составляли праймеры в соответствии с рекомендациями производителя (SEQ ID NO: 90-97). Подлежащая мутации плазмида составляет около 7000 п.н. Проверяли примерно 5 колоний на 1 подход путем секвенирования. Колонии, содержащие требуемые плазмиды, хранили в глицерине.
Проводили насыщающий мутагенез интегразы ВИЧ-1, получая большую комбинаторную библиотеку различных молекул интегразы ВИЧ-1. Методика была заимствована из Cornell et al. (Biochemistry, 57(5):604-613, 2018). Использовали несколько прямых праймеров, содержащих вырожденную последовательность NNS в мутационном сайте, и один обратный праймер в одной реакции ПЦР (SEQ ID NO: 90-97). Для получения мутантных молекул интегразы амплифицировали всю плазмиду. Праймеры оптимизировали для температуры плавления 68°C. Во время циклов температура отжига должна повышаться на 0,3°C за цикл. В таблице 8 приведен список аминокислотных мутаций.
Таблица 8. Сайты мутаций интегразы ВИЧ-1 в сравнении с аминокислотной последовательностью интегразы ВИЧ-1 дикого типа NC_001802.1 – NP_705928 (SEQ ID NO: 1)
аминокислота
Пример 14. Получение конструкций pRRLVPR для интегразы и проверка эффективности транкомплементации на клетках HEK293T
Создавали векторы pRRLIN, pRRLVPRIN и pRRLINGFP для применения при транскомплементации с VPR (таблица 9).
Таблица 9. Конструкции pRRL
Конструкции тестировали по экспрессии GFP. Клетки HEK293T трансфицировали pSICO MAXI, pSICO MINI и pRRL_INGFP для проверки эписомальной экспрессии pRRLINGFP. Экспрессия конструкции VPRINGFP в вырабатывающих лентивирус клетках оказалась положительной. Затем проверяли эффективность транскомплементации в клетках HEK293T.
Среду с LV ультрацентрифугировали, оставляли для ресуспендирования и засеивали клетки. Проводили инфицирование в объеме 0,6 мл (1,5×0,4). Добавляли полибрен. Определяли титры по цитометрии. Титры (1:100) представлены на фиг. 9.
Для сравнения модифицированных последовательностей интегразы при встраивании использовалась система транскомплементации с VPR.
Далее в примерах 15-19 создавали различные конструкции слитых белков с модифицированной гиперактивной транспозазой PiggyBac. Определяли активность общей и направленной транспозиции этих конструкций, получая соответствующие результаты, особенно для конструкций hCas9_mutated PB. Также получали данные по созданию и определению активности направленной транспозиции у конструкций слитых белков мутантных PB и ZFP. Проверяли различные линкеры, показывая, что XTEN обладает большей эффективностью, чем остальные исследованные линкеры. 5GGS и 7GGS также работали надлежащим образом, свидетельствуя, что длина линкера и его гибкость играют важную роль в его эффективности.
Пример 15. Способы получения слитых белков с модифицированными гиперактивными транспозазами PiggyBac и определение эффективности направленной транспозиции
Трансфекция
Клетки HEK293T высеивали накануне, чтобы достичь конфлюэнтности 70-80% в день трансфекции (обычно по 290 000 клеток в 12-луночном планшете). Трансфекцию проводили с помощью реагента липофектамин 3000 в соответствии с инструкциями производителя или PEI при соотношении ДНК-PEI = 1:3 в OptiMem.
Программируемую транспозазу (PT), направляющую РНК и плазмиды для транспозонов трансфицировали вместе в соотношении 1 PT:2,5 направляющая РНК:2,5 транспозона.
Клетки пассировали и поддерживали до требуемой конечной точки в зависимости от эксперимента.
Получение мутантов PB
Вводили различные мутации в последовательность hyPB, слитую с Cas9 (плазмида hCas9_PB), при помощи сайт-направленного мутагенеза в соответствии с инструкциями из набора для мутагенеза QuickChange Lightning Agilent. Составляли праймеры с помощью QuikChange Primer Design для получения следующих мутаций: PB R245A, PB R275-277A, PB R388A, PB S351A, PB W465A, PB R372A-K375A, PB D450N (SEQ ID NO: 100-106).
Активность Cas9
Плазмиду программируемой транспозазы с нуклеазой Cas9 и плазмиду для направляющей РНК трансфицировали вместе в соотношении 1:2,5. Через 48 ч собирали клетки и выделяли геномную ДНК. Проводили ПЦР с праймерами, нацеленными на 150-200 п.н. вокруг целевого сайта направляющей РНК (NGS-aavs fw и NGS-aavs rv, SEQ ID NO: 98-99). При второй реакции ПЦР вводили адаптеры и баркоды Illumina и проводили секвенирование MiSeq обычно в проточных кюветах 2x250 Nano. Результаты анализировали с помощью веб-инструмента CRISPR-GA.
Метод Genetrap
Получали транспозон RFP без промотора с акцептором сплайсинга перед ним и составляли направляющую РНК, нацеленные на PPR1-α и интрон-1 CD46, которые клонировали под управлением промотора U6. Флуоресценция RFP будет выявляться, только если транспозон случайно встроится в целевой участок или в район другого промотора. Для метода Genetrap клетки HEK293T трансфицировали транспозоном Genetrap, программируемой транспозазой и направляющей РНК, а сигналы RFP анализировали методом проточной цитометрии.
Репортерные линии клеток с расщепленным GFP
Получали репортерные линии клеток 293T для экспериментов по выявлению направленной транспозиции. Вкратце, эти линии клеток содержат целевую область (с разными последовательностями мишеней для направляющей РНК и ZFP) и последовательность акцептора сплайсинга, за которой следует половина кодирующей последовательности GFP. Эти линии клеток получали путем случайного встраивания репортерной кассеты с помощью гиперактивной версии транспозазы Sleeping Beauty, SB100X. При направленном введении транспозона с первой половиной последовательности GFP и с промотором и донором для сплайсинга появляются сигналы GFP, детектируемые методом проточной цитометрии.
Создавали и второй транспозон, содержащий половину последовательности GFP и полную последовательность RFP с конститутивным промотором EF1α перед ней для оценки направленности или случайности вставки. Примерно через 15 дней после трансфекции наблюдалось хорошее затухание эписомального сигнала, что позволяет отличить общую вставку (сигнал RFP) от направленной вставки (сигнал GFP).
Пример 16. Получение плазмидных конструкций для слитых белков с модифицированными гиперактивными транспозазами PiggyBac
Клонировали различные плазмидные конструкции для достижения слияния программируемого элемента, нацеленного на ДНК (Cas9, ZFN), и транспозазы млекопитающих (PiggyBac, SB100). Линкер между двумя модулями был вариабельным в различных конструкциях, его выбирали из библиотеки линкеров по SEQ ID NO: 50-63. Конструкции представлены в таблице 10.
Таблица 10. Список полученных слитых белков
Cas9 и hyPB
Cas9 и SB100
ZFN и hyPB
hCas9: оптимизированная по кодонам нуклеаза Cas9 человека; nCas9: оптимизированная по кодонам никаза Cas9 человека; dCas9: оптимизированная по кодонам «мертвая» Cas9 человека.
Пример 17. Эффективность транспозиции для различных линкеров
Клетки HEK293T трансфицировали конструкциями hCas9_PB с разными по длине и структуре линкерами (библиотека линкеров) и двумя разными направляющими РНК (AAVS1 № 1 и AAVS1 № 2). Через 48 ч после трансфекции выделяли геномную ДНК, амплифицировали целевую область методом ПЦР и секвенировали по методике секвенирования MiSeq фирмы Illumina.
Результаты. Конструкции с различными по длине и структуре линкерами не препятствуют активности нуклеазы Cas9. Линкер 4GGS дает большую активность Cas9 на сайтах-мишенях обеих направляющей РНК по сравнению с активностью hCas9 (фиг. 11).
Пример 18. Направленная транспозиция слитых белков с модифицированными гиперактивными транспозазами PiggyBac
18.1 GeneTrap
Определяли активность направленной транспозиции конструкции hCas9_PB (hCas9, соединенная с hyPB с помощью различных линкеров, описанных выше) при помощи транспозона Genetrap. Транспозон Genetrap содержит последовательность RFP без промотора с последовательностью акцептора сплайсинга перед ней, которая может экспрессироваться, только если она встроится в область промотора после донора сплайсинга.
Транспозон Genetrap трансфицировали вместе с направляющей РНК для интрона-1 PPR1 и программируемой транспозазой с различными конструкциями линкеров. Через 10 дней после трансфекции анализировали результаты по флуоресценции RFP методом проточной цитометрии.
Результаты. Направленная активность повышалась под действием программируемой транспозазы по сравнению со случайной вставкой hyPB, проявляя большую флуоресценцию при трансфекции программируемой транспозазой, чем при трансфекции hyPB дикого типа. Линкеры 8GGS, XTEN повышали направленную активность Genetrap по сравнению с другими линкерами (фиг. 12).
Репортерная линия клеток с расщепленным GFP
18.2 Направленная транспозиция hCas9_PB с различными линкерами
Определяли активность направленной транспозиции конструкции hCas9_PB при помощи репортерной линии клеток. Конструкции hCas9_PB с различными линкерами трансфецировали вместе с направляющей РНК для AAVS1 № 3 или TCR1α и половиной транспозона GFP. Результаты. Не отмечено больших отличий в отношении транспозиции конструкций с различными линкерами (фиг. 13).
18.3 Направленная транспозиция выбранных мутантов
Для дальнейших экспериментов по направленной транспозиции были выбраны PB 450 и PB 372-375-450 из-за хорошей эффективности их направленной транспозиции. Эксперименты проводили, как изложено выше, с использованием гРНК для AAVS1 № 3 и TCR1. Результаты. Направленная транспозиция hCas9_PB 450 и hCas9_PB 372-374-450 повышалась в 6-10 раз по сравнению с hCas9_PB с последовательностью hyPB дикого типа. При трансфекции hCas9 + hyPB в отдельных плазмидах проявлялась некоторая направленная активность, тогда как у hyPB без hCas9 проявлялась активность 0, свидетельствуя о том, что репортерные линии клеток с расщепленным GFP являются надежным методом направленной вставки для отбора таких вариантов, которые выполняют эту функцию выше уровня шума от других методов, которые недостаточно специфичны (фиг. 15).
18.4 Направленная и случайная транспозиция у выбранных мутантов PB
Направленную и случайную транспозицию оценивали при помощи двойного транспозона RFP-GFP, приведенного ранее для выбранных мутантов в примере 19.4. Красная флуоресценция означает вставку вообще (RFP экспрессируется конститутивно) примерно через 15 дней после трансфекции (что обеспечивает отсутствие эписомальных сигналов), а флуоресценция GFP означает направленную транспозицию. Результаты. Из фиг. 16 видно, что направленная транспозиция повышается по сравнению со случайной транспозицией у выбранных мутантов hCas9_PB D450N и hCas9_PB R372A K375A D450 в сравнении с hCas9_PB с последовательностью hyPB дикого типа. Общая эффективность транспозиции понижена у обоих мутантов, а направленные результаты согласуются с фиг. 15.
18.5 Направленная транспозиция конструкций ZFP-PB
Конструкции для слитых с гиперактивной PiggyBac цинк-пальцевых белков клонировали, используя нацеленную на ZFP последовательность TCR4, которая присутствует в репортерной линии клеток с расщепленным GFP, и hyPB либо hyPB с мутациями D450N. Клетки трансфицировали комбинациями ZFP-PB и ½ транспозона GFP по методике из Примера 15. Через 5 дней после трансфекции анализировали сигналы GFP. Результаты. У всех конструкций наблюдалась направленная транспозиция выше уровня фона (случайной вставки hyPB). Направленная транспозиция была выше у ZFP в N-концевом положении как для hyPB, так и для hyPB D450N (фиг. 18). Последовательность ZFP в этих экспериментах соответствует белку с 6 пальцевыми доменами с нуклеотидной и аминокислотной последовательностью по SEQ ID NO: 117 и 118, соответственно.
Далее в Примере 20 была разработана библиотека мутаций PB, которая была подвернута скринингу для идентификации модифицированной PB, положительной по направленной транспозиции. Были идентифицированы и подтверждены некоторые попадания для модифицированной PB, положительной по направленной транспозиции.
Пример 20. Получение библиотеки мутаций гиперактивной PiggyBac и скрининг на направленную транспозицию
Методы
Библиотека мутаций hyPB была разработана и приобретена у фирмы Twist Biosciences.
Таблица 11. Сайты мутаций для hyPiggyBac
Метод скрининга
Разработали метод скрининга для идентификации вариантов PiggyBac из полученной библиотеки мутантов, которые соединяются с целевым ДНК-связывающим белком типа Cas9 и выполняют определенные направленные транспозиции. Схема метода скрининга представлена на фиг. 19. Библиотеку PB клонировали методом сборки Golden Gate с помощью фермента Esp3I в лентивирусную плазмиду SIN для переноса, содержащую hCas9 и линкер XTEN, с последующими сайтами клонирования Esp3I перед NLS, получая слитый белок hCas9_XTEN_PB_NLS под управлением промотора CMV. После электропорации компетентных клеток ElectroMAX™ Stbl4™ фирмы Invitrogen получили около 6 000 000 колоний, из которых выделяли макси-препараты плазмид с помощью набора HiPure Maxiprep kit, LifeTechnologies. Получали лентивирусы (с помощью хелперных плазмид pMD2.G и psPAX2, приобретенных у Addgene) по методике получения лентивирусов фирмы Addgene. Лентивирусы ультрацентрифугировали и титровали методом кПЦР для анализа числа копий (с олигонуклеотидами SEQ ID NO: 107-110). Вкратце, за день до этого высеивали клетки HEK293T в 12-луночные планшеты. Клетки инфицировали лентивирусами с библиотекой и стандартным лентивирусом с GFP в разведениях 1/2, 1/10 для лентивирусов с библиотекой и 1/50, 1/100, 1/1000 для лентивирусов с GFP. Через 3 дня после заражения анализировали сигналы GFP методом проточной цитометрии. Собирали клетки и выделяли геномную ДНК. Проводили кПЦР для оценки числа копий гена WPRE и нормировали по числу копий гена РНКазы.
Репортерные клетки HEK293T инфицировали при MOI = 0,8 в квадратных чашках на 500 см2 с помощью полибрена 1:1000, а 10 млн. клеток засеивали накануне. Через 3-4 дня после заражения клетки трансфицировали плазмидой с направляющей РНК для AAVS1 по 8,1 пмоль и транспозоном с ½ GFP с помощью PEI 1:3. Накануне на чашки в 15 см высеивали 9 млн. клеток. Через 3-4 дня после трансфекции проводили сортировку клеток с помощью цитометра FACSAria с насадкой на 0,70 мкм. Контрольную трансфекцию проводили в чашках на 10 см, используя плазмиды для RFP и GFP при одинаковой молярности, и проводили анализ на цитометре Fortessa на наличие GFP-RFP-положительных клеток. После сортировки сразу выделяли геномную ДНК.
Для анализа мутантов PB, положительных по направленной транспозиции, использовали различные методы секвенирования.
Направленное секвенирование области PiggyBac в библиотеке
Область PiggyBac в 1116 п.н. у всех вариантов библиотеки подвергали ПЦР-амплификации с праймерами NGS cluster 1 fw и NGS cluster 2 rv, используя смесь KAPA HiFi Hotstart ReadyMix. При второй реакции ПЦР вводили адаптеры и баркоды Illumina, используя праймер NEBNext 9 и специальные баркоды Illumina (SEQ ID NO: 111-114). Направленное секвенирование проводили в проточных кюветах для MiSeq v2 или v3 фирмы Illumina. Праймер Index I7 заменяли на специальный праймер, обеспечивающий полное секвенирование различных вариантов.
Получение и секвенирование библиотеки последовательностей PiggyBac и Cas9 методом “дробовика”
Проводили ПЦР 6000 п.н. геномной ДНК из полученных при сортинге GFP-положительных клеток с праймерами CMV-F и SV40 pA rv (SEQ ID NO: 115 и 132), амплифицируя последовательность с Cas9 и PB с помощью смеси KAPA HiFi HotStart ReadyMix. Затем очищали ДНК с помощью набора Qiagen для экстракции гелей и фрагментировали до 500 п.н. с помощью Covaris S220 и микропробирок Crimp-Cap с волокном AFA. Библиотеку для “дробовика” готовили с помощью набора KAPA Hyperprep согласно инструкциям производителя.
Результаты
20.1 Получение библиотеки разнообразия hyPB
Репортерную линию клеток с ½ GFP инфицировали при MOI = 0,8 лентивирусами, содержащими hCas9_PB с мутациями в библиотеке PB. Через 3 дня после заражения клетки трансфицировали направляющей РНК для AAVS1 № 3 и ½ транспозона GFP при эффективности трансфекции 75-90%.
В первом эксперименте подвергали сортировке в целом 254 млн. клеток и получили 185 757 положительных клеток, то есть 0,073% положительных по направленной транспозиции вариантов. Во втором эксперименте подвергали сортировке 120 млн. клеток и получили 70 974 положительных клеток, то есть 0,059% положительных по направленной транспозиции вариантов (фиг. 21A и 21B).
Из положительных и отрицательных при сортировке клеток сразу выделяли геномную ДНК. ⅔ полученной ДНК подвергали обработке для направленного секвенирования, а ⅓ подвергали обработке для секвенирования библиотеки методом “дробовика”, как изложено выше в разделе «Методы» этого примера.
20.2 Скрининг библиотеки hyPB путем направленного секвенирования вариабельной области
Анализ положительных и отрицательных клеток с вариантами Cas9-PB проводили следующим образом. Картировали прочтения из направленного секвенирования по эталонной последовательности. Положения всех вариантов библиотеки получали при двух разных подходах: по положению, используя выровненные прочтения, и по последовательности, используя профили совпадения с окружающей последовательностью. Рассчитывали логарифмы кратности изменений подсчета у всех вариантов между положительными (GFP-положительные клетки с направленным встраиванием) и отрицательными образцами (образцы с ненаправленным встраиванием, независимо от того, происходило встраивание или нет) и отбирали лучшие варианты. Кроме того, проводили отбор отрицательных образцов со случайным встраиванием и при положительном отборе по RFP, где транспозон встраивается в геном случайным образом.
Результаты представлены на фиг. 22A-22K. Таким образом, используя самостоятельный высокопроизводительный метод скрининга комбинаторной библиотеки вариантов, была идентифицирована коллекция мутантов PiggyBack, способных выполнять сайт-направленное встраивание с высокой эффективностью, о чем свидетельствует их присутствие в популяции положительных, а не отрицательных клеток.
Далее проводили оценку направленной и случайной транспозиции у лучших положительных попаданий в повторе №1 с помощью двойного транспозона RFP-GFP, указанного ранее. Красная флуоресценция означает встраивание вообще (RFP экспрессируется конститутивно) примерно через 15 дней после трансфекции, а флуоресценция GFP означает направленную транспозицию.
Результаты. У варианта лучший 1 из повтора № 1 отмечалось повышение направленной транспозиции по сравнению с hCas9_PB и с hyPB дикого типа (фиг. 23A-23B). Проводилась независимая проверка направленности встраивания на нашей репортерной линии клеток, и наблюдалась значительная направленная активность по сравнению с версией дикого типа и с мутантом D450N.
20.3 Выявление избыточно представленных положительных попаданий
При скрининге было идентифицировано несколько положительных попаданий, чрезмерно представленных в популяции GPF по сравнению с отобранными отрицательными вариантами. Некоторые из них также не встречались в популяции RFP, представляющей вставки вообще, что означает повышение способности к встраиванию. Более того, RFP включает случайное и направленное встраивание. Таким образом, была идентифицирована коллекция комбинаторных мутантов PiggyBack, способных выполнять сайт-направленное встраивание с высокой эффективностью (фиг. 24A-24C).
20.4 Скрининг библиотеки hyPB путем секвенирования методом “дробовика”
Для секвенирования методом “дробовика” картировали прочтения по эталонной последовательности, проводили запрос вариантов с извлечением всех отклонений от эталона и рассчитывали евклидово и корреляционное расстояние между положительными и отрицательными показаниями аллелей. Наиболее отличающиеся позиции рассматривали как варианты и рассчитывали коэффициенты ассоциации между ними.
Результаты. Наряду с вариантами, включенными при разработке библиотеки, анализировали и варианты, введенные случайным образом под действием обратной транскриптазы лентивируса при создании вирусной библиотеки. Некоторые из этих новых вариантов связаны с положительными попаданиями и вероятно, что они выполняют направленное встраивание при комбинировании, возможно, они должны присутствовать в виде мутантов в версии варианта hyPB для выполнения направленного встраивания. Примеры – D450N и W465A на фиг. 25.
Мутантные последовательности PB, идентифицированные в примере 20, приведены в таблице 12 (SEQ ID NO: 120-129).
20.5 Проверка скрининга библиотеки hyPB
Проводили оценку направленной и случайной транспозиции некоторых комбинаций одиночных мутаций, отмеченных у лучший 1-1, идентифицированных при скрининге положительных попаданий (Unilarge-A, -B, -C и Unilarge-D), с помощью двойного транспозона RFP-GFP, указанного ранее. Красная флуоресценция означает встраивание вообще (RFP экспрессируется конститутивно) примерно через 15 дней после трансфекции, а флуоресценция GFP означает направленную транспозицию.
Результаты. Во всех случаях наблюдалось увеличение направленных вставок в сравнении со встраиванием вообще для Cas9, слитого с различными комбинациями мутантов hyPB с линкером 4GGS (Unilarge-A: D450N; Unilarge-B: R245A/D450N; Unilarge-C: R245A/G325A/D450N/S573P; Unilarge-D: R245A/G325A/S573P), по сравнению со слиянием Cas9 с версией hyPB дикого типа. Некоторые из исследованных комбинаций мутантов (Unilarge C: R245A/G325A/D450N/S573P) давали большое увеличение направленных вставок вплоть до 30% от общего числа случаев встраивания вместо 3% при слиянии с hyPB (фиг. 26).
Далее в Примере 21 представлен обзор по состоянию разработки различных дефектных по встраиванию вирусных векторов, а также по наилучшей системе транскомплементации; и данные по транскомплементации с помощью слитых белков IN.
Пример 21. Транскомплементация различных дефектных по интегразе систем
Для получения эффективной системы транскомлементации для тестирования слитых белков IN определяли эффективность продукции вируса и его способность к встраиванию путем трансфекции дефектного по встраиванию вируса и транскомплементированного вируса в клетки HEK293T и Jurkat при различных условиях. Клетки пассировали в течение 7 дней до тех пор, пока не проявлялось отсутствие эписомального сигнала, и анализировали сигналы GFP методом проточной цитометрии на 2-й, 5-й и 7-й день.
Результаты. Проявлялась различная эффективность продукции у разных систем, причем NILV был ближе всего к WT по продукции. Во всех случаях наблюдалось четкое восстановление встраивающей активности при проведении транскомплементации с помощью WT-HIV_IN (фиг. 27). Проверку попадания IN в систему транскомплементации проводили методом вестерн-блоттинга.
Таблица 12. Последовательности. “Н.к. последовательность” означает нуклеотидную последовательность, а “а.к. последовательность” – аминокислотную последовательность
с заменой D10K, E13K, D64A, D64E, G94D, G94E, G94R, G94K, D116A, D116E, N117D, N117E, N117R, N117K, S119A, S119P, S119T, S119G, S119D, S119E, S119R, S119K, N120D, N120E, N120R, N120K, T122K, T122I, T122V, T122A, T122R, A124D, A124E, A124R, A124K, A128T, E152A, E152D, Q168L, Q168A, E170G, F185K, R231G, R231K, R231D, R231E, R231S, K264R, K266R, K273R либо их комбинацией
с заменой G94D, G94E, G94R, G94K, N117D, N117E, N117R, N117K, S119A, S119P, S119T, S119G, S119D, S119E, S119R, S119K, N120D, N120E, N120R, N120K, A124D, A124E, A124R, A124K , R231G, R231K, R231D, R231E, R231K либо их комбинацией
с заменой G94D, G94E, G94R, G94K, N117D, N117E, N117R, N117K, S119A, S119P, S119T, S119G, S119D, S119E, S119R, S119K, N120D, N120E, N120R, N120K, T122K, T122I, T122V, T122A, T122R, A124D, A124E, A124R, A124K, R231G, R231K, R231D, R231E, R231S либо их комбинацией
с заменой K264R, K266R, K273R либо их комбинацией
с заменой D10K, E13K, D64A, D64E, D116A, D116E, A128T, E152A, E152D, Q168L, Q168A, E170G либо их комбинацией
с заменой Q168L и/или Q168A
с заменой R245A, D268N, R275A/R277A, K287A, K290A, K287A/K290A, R315A, G325A, R341A, D346N, N347A, N347S, T350A, S351E, S351P, S351A, K356E, N357A, R372A, K375A, R372A/A, R388A, K409A, K412A, K409A/K412A, K432A, D447A, D447N, D450N, R460A, K461A, R460A/K461A, W465A, S517A, T560A, S564P, S571N, S573A, K576A, H586A, I587A, M589V, S592G, F594L либо их комбинацией
с заменой D268N и/или D346N
с заменой K287A, K287A/K290A, R460A/K461A либо их комбинацией
с заменой S351E, S351P, S351A, K356E либо их комбинацией
с заменой T560A, S564P, S571N, S573A, M589V, S592G, F594L либо их комбинацией
с заменой G325A, N347A, N347S, T350A, W465A либо их комбинацией
с заменой K576A и/или I587A
с заменой H586A
с заменой R315A, R341A, R372A, K375A либо их комбинацией
Cas9 из Corynebacterium ulcerans
Cas9 из Corynebacterium diphtheria
Cas9 из Spiroplasma syrphidicola
Cas9 из Prevotella intermedia
Cas9 из Spiroplasma taiwanense
Cas9 из Streptococcus iniae
Cas9 из Belliella baltica
Cas9 из Psychroflexus torquisi
Cas9 из Streptococcus thermophilus
Cas9 из Listeria innocua
Cas9 из Campylobacter jejuni
Cas9 из Neisseria meningitidis
Cas9 из Streptococcus pyogenes
цинк-пальцевого домена 1
цинк-пальцевого домена 2
цинк-пальцевого домена 3
цинк-пальцевого домена 4
цинк-пальцевого домена 5
цинк-пальцевого домена 6
Cas9 человека (hCas9)
никазы Cas9 (nCas9)
«мертвой» Cas9 (dCas9)
гиперактивной транспозазы PiggyBac (PB)
гиперактивной транспозазы Sleeping Beauty (SB100)
Cas9 человека (hCas9)
никазы Cas9 (nCas9)
«мертвой» Cas9 (dCas9)
гиперактивной транспозазы PiggyBac (PB)
гиперактивной транспозазы Sleeping Beauty (SB100)
домена cPPT/CTS IN
с заменой R245A, R275A, R277A, R275A/R277A, G325A, N347A, N347S, S351E, S351P, S351A, R372A, K375A, R388A, D450N, W465A, T560A, S564P, S573A, M589V, S592G, F594L либо их комбинацией
с заменой на A в положении 245,
на R или A в положении 275,
на R или A в положении 277,
на A или G в положении 325,
на N или A в положении 347,
на E, P или A в положении 351,
на R в положении 372,
на A в положении 375,
на D или N в положении 450
на W или A в положении 465
на T или A в положении 560,
на P или S в положении 564,
на S или A в положении 573,
на G или S в положении 592,
на L или F в положении 594 либо их комбинацией
EEAFIDEVHEVQPTSSGSEILDEQNVIEQPGSSLASNRILTLPQR
TIRGKNKHCWSTSKPTRRSRVSALNIVRSQRGPTRMCRNIYDP
LLCFKLFFTDEIISEIVKWTNAEISLKRRESMTSATFRDTNEDEI
YAFFGILVMTAVRKDNHMSTDDLFDRSLSMVYVSVMSRDRF
DFLIRCLRMDDKSIRPTLRENDVFTPVAKIWDLFIHQCIQNYTP
GAHLTIDEQLLGFRGRCPFRVYIPNKPSKYGIKILMMCDSGTK
YMINGMPYLGRGTQTNGVPLAEYYVKELSKPVHGSCRNITCD
NWFTEIPLAKNLLQEPYKLTIVGTVRSNAREIPEVLKNSRSRPV
GTSMFCFDGPLTLVSYKPKPAKMVYLLSSCDEDASINESTGKP
QMVMYYNQTKGGVDTL(D/N)QMCSVMTCSRKTNR(W/A)PM
ALLYGMINIACINSFIIYSHNVSSKGEKVQSRKKFMRNLYMGL
TSSFMRKRLEAPTLKRYLRDNISNILPKEVPGTSDDSTEEPVMK
KRTYCTYCPPKIRRKASASCKKCKKVICREHNIDMCQGCL
причем:
в положении 450 может быть D или N
в положении 465 может быть W или A
VQSDTEEAFIDEVHEVQPTSSGSEILDEQNVIEQPGSSLA
SNRILTLPQRTIRGKNKHCWSTSKPTRRSRVSALNIVRSQ
RGPTRMCRNIYDPLLCFKLFFTDEIISEIVKWTNAEISLK
RRESMTSATFRDTNEDEIYAFFGILVMTAVRKDNHMST
DDLFDRSLSMVYVSVMSRDRFDFLIRCLRMDDKSIRPTL
RENDVFTPVAKIWDLFIHQCIQNYTPGAHLTIDEQLLGF
AGACPFRVYIPNKPSKYGIKILMMCDSGTKYMINGMPY
LGRGTQTNGVPLGEYYVKELSKPVHGSCRNITCDAWFT
PIPLAKNLLQEPYKLTIVGTVRSNAREIPEVLKNSRSRPV
GTSMFCFDGPLTLVSYKPKPAKMVYLLSSCDEDASINES
TGKPQMVMYYNQTKGGVDTL(D/N)QMCSVMTCSRKT
NR(W/A)PMALLYGMINIACINSFIIYSHNVSSKGEKVQSR
KKFMRNLYMGLTSSFMRKRLEAPTLKRYLRDNISNILP
KEVPGTSDDSTEEPVMKKRTYCAYCPSKIRRKASASCK
KCKKVICREHNIDMCQSCF
причем:
в положении 450 может быть D или N
в положении 465 может быть W или A
VQSDTEEAFIDEVHEVQPTSSGSEILDEQNVIEQPGSSLA
SNRILTLPQRTIRGKNKHCWSTSKPTRRSRVSALNIVRSQ
RGPTRMCRNIYDPLLCFKLFFTDEIISEIVKWTNAEISLK
RRESMTSATFRDTNEDEIYAFFGILVMTAVRKDNHMST
DDLFDRSLSMVYVSVMSRDRFDFLIRCLRMDDKSIRPTL
RENDVFTPVAKIWDLFIHQCIQNYTPGAHLTIDEQLLGF
RGRCPFRVYIPNKPSKYGIKILMMCDSGTKYMINGMPY
LGRGTQTNGVPLAEYYVKELSKPVHGSCRNITCDAWFT
AIPLAKNLLQEPYKLTIVGTVRSNAREIPEVLKNSRSRPV
GTSMFCFDGPLTLVSYKPKPAKMVYLLSSCDEDASINES
TGKPQMVMYYNQTKGGVDTL(D/N)QMCSVMTCSRKT
NR(W/A)PMALLYGMINIACINSFIIYSHNVSSKGEKVQSR
KKFMRNLYMGLTSSFMRKRLEAPTLKRYLRDNISNILP
KEVPGTSDDSTEEPVMKKRTYCAYCPSKIRRKASAACK
KCKKVICREHNIDMCQSCF
причем:
в положении 450 может быть D или N
в положении 465 может быть W или A
VQSDTEEAFIDEVHEVQPTSSGSEILDEQNVIEQPGSSLA
SNRILTLPQRTIRGKNKHCWSTSKPTRRSRVSALNIVRSQ
RGPTRMCRNIYDPLLCFKLFFTDEIISEIVKWTNAEISLK
RRESMTSATFRDTNEDEIYAFFGILVMTAVRKDNHMST
DDLFDRSLSMVYVSVMSRDRFDFLIRCLRMDDKSIRPTL
RENDVFTPVRKIWDLFIHQCIQNYTPGAHLTIDEQLLGF
RGRCPFRVYIPNKPSKYGIKILMMCDSGTKYMINGMPY
LGRGTQTNGVPLGEYYVKELSKPVHGSCRNITCDAWFT
SIPLAKNLLQEPYKLTIVGTVASNKREIPEVLKNSRSRPV
GTSMFCFDGPLTLVSYKPKPAKMVYLLSSCDEDASINES
TGKPQMVMYYNQTKGGVDTL(D/N)QMCSVMTCSRKT
NR(W/A)PMALLYGMINIACINSFIIYSHNVSSKGEKVQSR
KKFMRNLYMGLTSSFMRKRLEAPTLKRYLRDNISNILP
KEVPGTSDDSTEEPVMKKRTYCTYCPSKIRRKASASCK
KCKKVICREHNIDMCQSCF
причем:
в положении 450 может быть D или N
в положении 465 может быть W или A
VQSDTEEAFIDEVHEVQPTSSGSEILDEQNVIEQPGSSLA
SNRILTLPQRTIRGKNKHCWSTSKPTRRSRVSALNIVRSQ
RGPTRMCRNIYDPLLCFKLFFTDEIISEIVKWTNAEISLK
RRESMTSATFRDTNEDEIYAFFGILVMTAVRKDNHMST
DDLFDRSLSMVYVSVMSRDRFDFLIRCLRMDDKSIRPTL
RENDVFTPVRKIWDLFIHQCIQNYTPGAHLTIDEQLLGF
RGRCPFRVYIPNKPSKYGIKILMMCDSGTKYMINGMPY
LGRGTQTNGVPLGEYYVKELSKPVHGSCRNITCDNWFT
SIPLAKNLLQEPYKLTIVGTVRSNKREIPEVLKNSRSRPV
GTSMFCFDGPLTLVSYKPKPAKMVYLLSSCDEDASINES
TGKPQMVMYYNQTKGGVDTL(D/N)QMCSVMTCSRKT
NR(W/A)PMALLYGMINIACINSFIIYSHNVSSKGEKVQSR
KKFMRNLYMGLTSSFMRKRLEAPTLKRYLRDNISNILP
KEVPGTSDDSTEEPVMKKRTYCTYCPSKIRRKASASCK
KCKKVICREHNIDMCQGCF
причем:
в положении 450 может быть D или N
в положении 465 может быть W или A
VQSDTEEAFIDEVHEVQPTSSGSEILDEQNVIEQPGSSLA
SNRILTLPQRTIRGKNKHCWSTSKPTRRSRVSALNIVRSQ
RGPTRMCRNIYDPLLCFKLFFTDEIISEIVKWTNAEISLK
RRESMTSATFRDTNEDEIYAFFGILVMTAVRKDNHMST
DDLFDRSLSMVYVSVMSRDRFDFLIRCLRMDDKSIRPTL
RENDVFTPVRKIWDLFIHQCIQNYTPGAHLTIDEQLLGF
RGRCPFRVYIPNKPSKYGIKILMMCDSGTKYMINGMPY
LGRGTQTNGVPLGEYYVKELSKPVHGSCRNITCDNWFT
SIPLAKNLLQEPYKLTIVGTVRSNKREIPEVLKNSRSRPV
GTSMFCFDGPLTLVSYKPKPAKMVYLLSSCDEDASINES
TGKPQMVMYYNQTKGGVDTL(D/N)QMCSVMTCSRKT
NR(W/A)PMALLYGMINIACINSFIIYSHNVSSKGEKVQSR
KKFMRNLYMGLTSSFMRKRLEAPTLKRYLRDNISNILP
KEVPGTSDDSTEEPVMKKRTYCTYCPSKIRRKASASCK
KCKKVICREHNIDMCQSCF
причем:
в положении 450 может быть D или N
в положении 465 может быть W или A
VQSDTEEAFIDEVHEVQPTSSGSEILDEQNVIEQPGSSLA
SNRILTLPQRTIRGKNKHCWSTSKPTRRSRVSALNIVRSQ
RGPTRMCRNIYDPLLCFKLFFTDEIISEIVKWTNAEISLK
RRESMTSATFRDTNEDEIYAFFGILVMTAVRKDNHMST
DDLFDRSLSMVYVSVMSRDRFDFLIRCLRMDDKSIRPTL
RENDVFTPVAKIWDLFIHQCIQNYTPGAHLTIDEQLLGF
AGACPFRVYIPNKPSKYGIKILMMCDSGTKYMINGMPY
LGRGTQTNGVPLGEYYVKELSKPVHGSCRNITCDNWFT
SIPLAKNLLQEPYKLTIVGTVASNAREIPEVLKNSRSRPV
GTSMFCFDGPLTLVSYKPKPAKMVYLLSSCDEDASINES
TGKPQMVMYYNQTKGGVDTL(D/N)QMCSVMTCSRKT
NR(W/A)PMALLYGMINIACINSFIIYSHNVSSKGEKVQSR
KKFMRNLYMGLTSSFMRKRLEAPTLKRYLRDNISNILP
KEVPGTSDDSTEEPVMKKRTYCTYCPSKIRRKASASCK
KCKKVICREHNIDMCQSCL
причем:
в положении 450 может быть D или N
в положении 465 может быть W или A
VQSDTEEAFIDEVHEVQPTSSGSEILDEQNVIEQPGSSLA
SNRILTLPQRTIRGKNKHCWSTSKPTRRSRVSALNIVRSQ
RGPTRMCRNIYDPLLCFKLFFTDEIISEIVKWTNAEISLK
RRESMTSATFRDTNEDEIYAFFGILVMTAVRKDNHMST
DDLFDRSLSMVYVSVMSRDRFDFLIRCLRMDDKSIRPTL
RENDVFTPVRKIWDLFIHQCIQNYTPGAHLTIDEQLLGF
AGRCPFRVYIPNKPSKYGIKILMMCDSGTKYMINGMPY
LGRGTQTNGVPLAEYYVKELSKPVHGSCRNITCDSWFT
AIPLAKNLLQEPYKLTIVGTVASNKREIPEVLKNSRSRPV
GTSMFCFDGPLTLVSYKPKPAKMVYLLSSCDEDASINES
TGKPQMVMYYNQTKGGVDTL(D/N)QMCSVMTCSRKT
NR(W/A)PMALLYGMINIACINSFIIYSHNVSSKGEKVQSR
KKFMRNLYMGLTSSFMRKRLEAPTLKRYLRDNISNILP
KEVPGTSDDSTEEPVMKKRTYCAYCPSKIRRKASASCK
KCKKVICREHNIDMCQGCF
причем:
в положении 450 может быть D или N
в положении 465 может быть W или A
VQSDTEEAFIDEVHEVQPTSSGSEILDEQNVIEQPGSSLA
SNRILTLPQRTIRGKNKHCWSTSKPTRRSRVSALNIVRSQ
RGPTRMCRNIYDPLLCFKLFFTDEIISEIVKWTNAEISLK
RRESMTSATFRDTNEDEIYAFFGILVMTAVRKDNHMST
DDLFDRSLSMVYVSVMSRDRFDFLIRCLRMDDKSIRPTL
RENDVFTPVRKIWDLFIHQCIQNYTPGAHLTIDEQLLGF
AGRCPFRVYIPNKPSKYGIKILMMCDSGTKYMINGMPY
LGRGTQTNGVPLGEYYVKELSKPVHGSCRNITCDNWFT
AIPLAKNLLQEPYKLTIVGTVASNAREIPEVLKNSRSRPV
GTSMFCFDGPLTLVSYKPKPAKMVYLLSSCDEDASINES
TGKPQMVMYYNQTKGGVDTL(D/N)QMCSVMTCSRKT
NR(W/A)PMALLYGMINIACINSFIIYSHNVSSKGEKVQSR
KKFMRNLYMGLTSSFMRKRLEAPTLKRYLRDNISNILP
KEVPGTSDDSTEEPVMKKRTYCAYCPSKIRRKASASCK
KCKKVICREHNIDMCQGCF
причем:
в положении 450 может быть D или N
в положении 465 может быть W или A
n – целое число от 1 до 50
n – целое число от 1 до 50
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> УНИВЕРСИТАТ ПОМПЕУ ФАБРА
<120> TARGETED GENE EDITING CONSTRUCTS AND METHODS OF USING THE SAME
<130> 4349.001PC01/TJS/JCA/LXR
<150> US 62/860,186
<151> 2019-06-11
<160> 134
<170> PatentIn version 3.5
<210> 1
<211> 288
<212> PRT
<213> Artificial Sequence
<220>
<223> Wildtype HIV-1 integrase amino acid sequence
<400> 1
Phe Leu Asp Gly Ile Asp Lys Ala Gln Asp Glu His Glu Lys Tyr His
1 5 10 15
Ser Asn Trp Arg Ala Met Ala Ser Asp Phe Asn Leu Pro Pro Val Val
20 25 30
Ala Lys Glu Ile Val Ala Ser Cys Asp Lys Cys Gln Leu Lys Gly Glu
35 40 45
Ala Met His Gly Gln Val Asp Cys Ser Pro Gly Ile Trp Gln Leu Asp
50 55 60
Cys Thr His Leu Glu Gly Lys Val Ile Leu Val Ala Val His Val Ala
65 70 75 80
Ser Gly Tyr Ile Glu Ala Glu Val Ile Pro Ala Glu Thr Gly Gln Glu
85 90 95
Thr Ala Tyr Phe Leu Leu Lys Leu Ala Gly Arg Trp Pro Val Lys Thr
100 105 110
Ile His Thr Asp Asn Gly Ser Asn Phe Thr Gly Ala Thr Val Arg Ala
115 120 125
Ala Cys Trp Trp Ala Gly Ile Lys Gln Glu Phe Gly Ile Pro Tyr Asn
130 135 140
Pro Gln Ser Gln Gly Val Val Glu Ser Met Asn Lys Glu Leu Lys Lys
145 150 155 160
Ile Ile Gly Gln Val Arg Asp Gln Ala Glu His Leu Lys Thr Ala Val
165 170 175
Gln Met Ala Val Phe Ile His Asn Phe Lys Arg Lys Gly Gly Ile Gly
180 185 190
Gly Tyr Ser Ala Gly Glu Arg Ile Val Asp Ile Ile Ala Thr Asp Ile
195 200 205
Gln Thr Lys Glu Leu Gln Lys Gln Ile Thr Lys Ile Gln Asn Phe Arg
210 215 220
Val Tyr Tyr Arg Asp Ser Arg Asn Pro Leu Trp Lys Gly Pro Ala Lys
225 230 235 240
Leu Leu Trp Lys Gly Glu Gly Ala Val Val Ile Gln Asp Asn Ser Asp
245 250 255
Ile Lys Val Val Pro Arg Arg Lys Ala Lys Ile Ile Arg Asp Tyr Gly
260 265 270
Lys Gln Met Ala Gly Asp Asp Cys Val Ala Ser Arg Gln Asp Glu Asp
275 280 285
<210> 2
<211> 867
<212> DNA
<213> Artificial Sequence
<220>
<223> Wildtype HIV-1 integrase nucleic acid sequence
<400> 2
tttttagatg gaatagataa ggcccaagat gaacatgaga aatatcacag taattggaga 60
gcaatggcta gtgattttaa cctgccacct gtagtagcaa aagaaatagt agccagctgt 120
gataaatgtc agctaaaagg agaagccatg catggacaag tagactgtag tccaggaata 180
tggcaactag attgtacaca tttagaagga aaagttatcc tggtagcagt tcatgtagcc 240
agtggatata tagaagcaga agttattcca gcagaaacag ggcaggaaac agcatatttt 300
cttttaaaat tagcaggaag atggccagta aaaacaatac atactgacaa tggcagcaat 360
ttcaccggtg ctacggttag ggccgcctgt tggtgggcgg gaatcaagca ggaatttgga 420
attccctaca atccccaaag tcaaggagta gtagaatcta tgaataaaga attaaagaaa 480
attataggac aggtaagaga tcaggctgaa catcttaaga cagcagtaca aatggcagta 540
ttcatccaca attttaaaag aaaagggggg attggggggt acagtgcagg ggaaagaata 600
gtagacataa tagcaacaga catacaaact aaagaattac aaaaacaaat tacaaaaatt 660
caaaattttc gggtttatta cagggacagc agaaatccac tttggaaagg accagcaaag 720
ctcctctgga aaggtgaagg ggcagtagta atacaagata atagtgacat aaaagtagtg 780
ccaagaagaa aagcaaagat cattagggat tatggaaaac agatggcagg tgatgattgt 840
gtggcaagta gacaggatga ggattag 867
<210> 3
<211> 288
<212> PRT
<213> Artificial Sequence
<220>
<223> Modified HIV-1 integrase
<220>
<221> misc_feature
<222> (10)..(10)
<223> Xaa can be Lys, Asp
<220>
<221> misc_feature
<222> (11)..(11)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (13)..(13)
<223> Xaa can be Glu, Lys
<220>
<221> misc_feature
<222> (64)..(64)
<223> Xaa can be Ala, Glu, Asp
<220>
<221> misc_feature
<222> (94)..(94)
<223> Xaa can be Asp, Glu, Arg, Lys, Gly
<220>
<221> misc_feature
<222> (116)..(116)
<223> Xaa can be Asp, Ala, Glu
<220>
<221> misc_feature
<222> (117)..(117)
<223> Xaa can be Asp, Glu, Arg, Lys, Asn
<220>
<221> misc_feature
<222> (119)..(119)
<223> Xaa can be Ala, Pro, Thr, Gly, Asp, Glu, Arg, Lys, Ser
<220>
<221> misc_feature
<222> (120)..(120)
<223> Xaa can be Asp, Glu, Arg, Lys, Asn
<220>
<221> misc_feature
<222> (122)..(122)
<223> Xaa can be Lys, Ile, Val, Ala, Arg, Thr
<220>
<221> misc_feature
<222> (124)..(124)
<223> Xaa can be Asp, Glu, Arg, Lys, Ala
<220>
<221> misc_feature
<222> (125)..(125)
<223> Xaa can be Asp, Glu, Arg, Lys, Thr
<220>
<221> misc_feature
<222> (128)..(128)
<223> Xaa can be Ala, Thr
<220>
<221> misc_feature
<222> (152)..(152)
<223> Xaa can be Ala, Asp, Glu
<220>
<221> misc_feature
<222> (168)..(168)
<223> Xaa can be Leu, Ala, Gln
<220>
<221> misc_feature
<222> (170)..(170)
<223> Xaa can be Glu, Gly
<220>
<221> misc_feature
<222> (185)..(185)
<223> Xaa can be Phe, Lys
<220>
<221> misc_feature
<222> (231)..(231)
<223> Xaa can be Arg, Gly, Lys, Asp, Glu, Ser
<220>
<221> misc_feature
<222> (264)..(264)
<223> Xaa can be Lys, Arg
<220>
<221> misc_feature
<222> (266)..(266)
<223> Xaa can be Lys, Arg
<220>
<221> misc_feature
<222> (273)..(273)
<223> Xaa can be Lys, Arg
<400> 3
Phe Leu Asp Gly Ile Asp Lys Ala Gln Xaa Xaa His Xaa Lys Tyr His
1 5 10 15
Ser Asn Trp Arg Ala Met Ala Ser Asp Phe Asn Leu Pro Pro Val Val
20 25 30
Ala Lys Glu Ile Val Ala Ser Cys Asp Lys Cys Gln Leu Lys Gly Glu
35 40 45
Ala Met His Gly Gln Val Asp Cys Ser Pro Gly Ile Trp Gln Leu Xaa
50 55 60
Cys Thr His Leu Glu Gly Lys Val Ile Leu Val Ala Val His Val Ala
65 70 75 80
Ser Gly Tyr Ile Glu Ala Glu Val Ile Pro Ala Glu Thr Xaa Gln Glu
85 90 95
Thr Ala Tyr Phe Leu Leu Lys Leu Ala Gly Arg Trp Pro Val Lys Thr
100 105 110
Ile His Thr Xaa Xaa Gly Xaa Xaa Phe Xaa Gly Xaa Xaa Val Arg Xaa
115 120 125
Ala Cys Trp Trp Ala Gly Ile Lys Gln Glu Phe Gly Ile Pro Tyr Asn
130 135 140
Pro Gln Ser Gln Gly Val Val Xaa Ser Met Asn Lys Glu Leu Lys Lys
145 150 155 160
Ile Ile Gly Gln Val Arg Asp Xaa Ala Glu His Leu Lys Thr Ala Val
165 170 175
Gln Met Ala Val Phe Ile His Asn Xaa Lys Arg Lys Gly Gly Ile Gly
180 185 190
Gly Tyr Ser Ala Gly Glu Arg Ile Val Asp Ile Ile Ala Thr Asp Ile
195 200 205
Gln Thr Lys Glu Leu Gln Lys Gln Ile Thr Lys Ile Gln Asn Phe Arg
210 215 220
Val Tyr Tyr Arg Asp Ser Xaa Asn Pro Leu Trp Lys Gly Pro Ala Lys
225 230 235 240
Leu Leu Trp Lys Gly Glu Gly Ala Val Val Ile Gln Asp Asn Ser Asp
245 250 255
Ile Lys Val Val Pro Arg Arg Xaa Ala Xaa Ile Ile Arg Asp Tyr Gly
260 265 270
Xaa Gln Met Ala Gly Asp Asp Cys Val Ala Ser Arg Gln Asp Glu Asp
275 280 285
<210> 4
<211> 288
<212> PRT
<213> Artificial Sequence
<220>
<223> Modified integrase with impaired DNA binding
<220>
<221> misc_feature
<222> (94)..(94)
<223> Xaa can be Gly, Asp, Glu, Arg, Lys
<220>
<221> misc_feature
<222> (117)..(117)
<223> Xaa can be Asn, Asp, Glu, Arg, Lys
<220>
<221> misc_feature
<222> (119)..(119)
<223> Xaa can be Ser, Ala, Pro, Thr, Gly, Asp, Glu, Arg, Lys
<220>
<221> misc_feature
<222> (120)..(120)
<223> Xaa can be Asn, Asp, Glu, Arg, Lys
<220>
<221> misc_feature
<222> (124)..(124)
<223> Xaa can be Asp, Glu, Arg, Lys
<220>
<221> misc_feature
<222> (125)..(125)
<223> Xaa can be Asp, Glu, Arg, Lys, Thr
<220>
<221> misc_feature
<222> (231)..(231)
<223> Xaa can be Arg, Gly, Lys, Asp, Glu, Ser
<400> 4
Phe Leu Asp Gly Ile Asp Lys Ala Gln Asp Glu His Glu Lys Tyr His
1 5 10 15
Ser Asn Trp Arg Ala Met Ala Ser Asp Phe Asn Leu Pro Pro Val Val
20 25 30
Ala Lys Glu Ile Val Ala Ser Cys Asp Lys Cys Gln Leu Lys Gly Glu
35 40 45
Ala Met His Gly Gln Val Asp Cys Ser Pro Gly Ile Trp Gln Leu Asp
50 55 60
Cys Thr His Leu Glu Gly Lys Val Ile Leu Val Ala Val His Val Ala
65 70 75 80
Ser Gly Tyr Ile Glu Ala Glu Val Ile Pro Ala Glu Thr Xaa Gln Glu
85 90 95
Thr Ala Tyr Phe Leu Leu Lys Leu Ala Gly Arg Trp Pro Val Lys Thr
100 105 110
Ile His Thr Asp Xaa Gly Xaa Xaa Phe Thr Gly Xaa Xaa Val Arg Ala
115 120 125
Ala Cys Trp Trp Ala Gly Ile Lys Gln Glu Phe Gly Ile Pro Tyr Asn
130 135 140
Pro Gln Ser Gln Gly Val Val Glu Ser Met Asn Lys Glu Leu Lys Lys
145 150 155 160
Ile Ile Gly Gln Val Arg Asp Gln Ala Glu His Leu Lys Thr Ala Val
165 170 175
Gln Met Ala Val Phe Ile His Asn Phe Lys Arg Lys Gly Gly Ile Gly
180 185 190
Gly Tyr Ser Ala Gly Glu Arg Ile Val Asp Ile Ile Ala Thr Asp Ile
195 200 205
Gln Thr Lys Glu Leu Gln Lys Gln Ile Thr Lys Ile Gln Asn Phe Arg
210 215 220
Val Tyr Tyr Arg Asp Ser Xaa Asn Pro Leu Trp Lys Gly Pro Ala Lys
225 230 235 240
Leu Leu Trp Lys Gly Glu Gly Ala Val Val Ile Gln Asp Asn Ser Asp
245 250 255
Ile Lys Val Val Pro Arg Arg Lys Ala Lys Ile Ile Arg Asp Tyr Gly
260 265 270
Lys Gln Met Ala Gly Asp Asp Cys Val Ala Ser Arg Gln Asp Glu Asp
275 280 285
<210> 5
<211> 288
<212> PRT
<213> Artificial Sequence
<220>
<223> Modified integrase with enhanced DNA binding
<220>
<221> Misc_feature
<222> (94)..(94)
<223> Xaa can be Gly, Asp, Glu, Arg, Lys
<220>
<221> Misc_feature
<222> (117)..(117)
<223> Xaa can be Asn, Asp, Glu, Arg, Lys
<220>
<221> Misc_feature
<222> (119)..(119)
<223> Xaa can be Ala, Pro, Thr, Gly, Asp, Glu, Arg, Lys, Ser
<220>
<221> Misc_feature
<222> (120)..(120)
<223> Xaa can be Asn, Asp, Glu, Arg, Lys
<220>
<221> Misc_feature
<222> (122)..(122)
<223> Xaa can be Thr, Lys, Ile, Val, Ala, Arg
<220>
<221> Misc_feature
<222> (124)..(124)
<223> Xaa can be Asp, Glu, Arg, Lys, Ala
<220>
<221> Misc_feature
<222> (125)..(125)
<223> Xaa can be Asp, Glu, Arg, Lys, Thr
<220>
<221> Misc_feature
<222> (168)..(168)
<223> Xaa can be Leu, Ala, Gln
<220>
<221> Misc_feature
<222> (231)..(231)
<223> Xaa can be Arg, Gly, Lys, Asp, Glu, Ser
<400> 5
Phe Leu Asp Gly Ile Asp Lys Ala Gln Asp Glu His Glu Lys Tyr His
1 5 10 15
Ser Asn Trp Arg Ala Met Ala Ser Asp Phe Asn Leu Pro Pro Val Val
20 25 30
Ala Lys Glu Ile Val Ala Ser Cys Asp Lys Cys Gln Leu Lys Gly Glu
35 40 45
Ala Met His Gly Gln Val Asp Cys Ser Pro Gly Ile Trp Gln Leu Asp
50 55 60
Cys Thr His Leu Glu Gly Lys Val Ile Leu Val Ala Val His Val Ala
65 70 75 80
Ser Gly Tyr Ile Glu Ala Glu Val Ile Pro Ala Glu Thr Xaa Gln Glu
85 90 95
Thr Ala Tyr Phe Leu Leu Lys Leu Ala Gly Arg Trp Pro Val Lys Thr
100 105 110
Ile His Thr Asp Xaa Gly Xaa Xaa Phe Xaa Gly Xaa Xaa Val Arg Ala
115 120 125
Ala Cys Trp Trp Ala Gly Ile Lys Gln Glu Phe Gly Ile Pro Tyr Asn
130 135 140
Pro Gln Ser Gln Gly Val Val Glu Ser Met Asn Lys Glu Leu Lys Lys
145 150 155 160
Ile Ile Gly Gln Val Arg Asp Xaa Ala Glu His Leu Lys Thr Ala Val
165 170 175
Gln Met Ala Val Phe Ile His Asn Phe Lys Arg Lys Gly Gly Ile Gly
180 185 190
Gly Tyr Ser Ala Gly Glu Arg Ile Val Asp Ile Ile Ala Thr Asp Ile
195 200 205
Gln Thr Lys Glu Leu Gln Lys Gln Ile Thr Lys Ile Gln Asn Phe Arg
210 215 220
Val Tyr Tyr Arg Asp Ser Xaa Asn Pro Leu Trp Lys Gly Pro Ala Lys
225 230 235 240
Leu Leu Trp Lys Gly Glu Gly Ala Val Val Ile Gln Asp Asn Ser Asp
245 250 255
Ile Lys Val Val Pro Arg Arg Lys Ala Lys Ile Ile Arg Asp Tyr Gly
260 265 270
Lys Gln Met Ala Gly Asp Asp Cys Val Ala Ser Arg Gln Asp Glu Asp
275 280 285
<210> 6
<211> 288
<212> PRT
<213> Artificial Sequence
<220>
<223> Modified integrase with acetylation mutations
<220>
<221> misc_feature
<222> (264)..(264)
<223> Xaa can be Lys, Arg
<220>
<221> misc_feature
<222> (266)..(266)
<223> Xaa can be Lys, Arg
<220>
<221> misc_feature
<222> (273)..(273)
<223> Xaa can be Lys, Arg
<400> 6
Phe Leu Asp Gly Ile Asp Lys Ala Gln Asp Glu His Glu Lys Tyr His
1 5 10 15
Ser Asn Trp Arg Ala Met Ala Ser Asp Phe Asn Leu Pro Pro Val Val
20 25 30
Ala Lys Glu Ile Val Ala Ser Cys Asp Lys Cys Gln Leu Lys Gly Glu
35 40 45
Ala Met His Gly Gln Val Asp Cys Ser Pro Gly Ile Trp Gln Leu Asp
50 55 60
Cys Thr His Leu Glu Gly Lys Val Ile Leu Val Ala Val His Val Ala
65 70 75 80
Ser Gly Tyr Ile Glu Ala Glu Val Ile Pro Ala Glu Thr Gly Gln Glu
85 90 95
Thr Ala Tyr Phe Leu Leu Lys Leu Ala Gly Arg Trp Pro Val Lys Thr
100 105 110
Ile His Thr Asp Asn Gly Ser Asn Phe Thr Gly Ala Thr Val Arg Ala
115 120 125
Ala Cys Trp Trp Ala Gly Ile Lys Gln Glu Phe Gly Ile Pro Tyr Asn
130 135 140
Pro Gln Ser Gln Gly Val Val Glu Ser Met Asn Lys Glu Leu Lys Lys
145 150 155 160
Ile Ile Gly Gln Val Arg Asp Gln Ala Glu His Leu Lys Thr Ala Val
165 170 175
Gln Met Ala Val Phe Ile His Asn Phe Lys Arg Lys Gly Gly Ile Gly
180 185 190
Gly Tyr Ser Ala Gly Glu Arg Ile Val Asp Ile Ile Ala Thr Asp Ile
195 200 205
Gln Thr Lys Glu Leu Gln Lys Gln Ile Thr Lys Ile Gln Asn Phe Arg
210 215 220
Val Tyr Tyr Arg Asp Ser Arg Asn Pro Leu Trp Lys Gly Pro Ala Lys
225 230 235 240
Leu Leu Trp Lys Gly Glu Gly Ala Val Val Ile Gln Asp Asn Ser Asp
245 250 255
Ile Lys Val Val Pro Arg Arg Xaa Ala Xaa Ile Ile Arg Asp Tyr Gly
260 265 270
Xaa Gln Met Ala Gly Asp Asp Cys Val Ala Ser Arg Gln Asp Glu Asp
275 280 285
<210> 7
<211> 288
<212> PRT
<213> Artificial Sequence
<220>
<223> Modified integrase with mutations in retroviral integrative
recombination
<220>
<221> misc_feature
<222> (10)..(10)
<223> Xaa can be Asp, Lys
<220>
<221> misc_feature
<222> (11)..(11)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (13)..(13)
<223> Xaa can be Glu, Lys
<220>
<221> misc_feature
<222> (64)..(64)
<223> Xaa can be Asp, Ala, Glu
<220>
<221> misc_feature
<222> (116)..(116)
<223> Xaa can be Ala, Glu, Asp
<220>
<221> misc_feature
<222> (128)..(128)
<223> Xaa can be Ala, Thr
<220>
<221> misc_feature
<222> (152)..(152)
<223> Xaa can be Glu, Ala, Asp
<220>
<221> misc_feature
<222> (168)..(168)
<223> Xaa can be Gln, Leu, Ala
<220>
<221> misc_feature
<222> (170)..(170)
<223> Xaa can be Asp, Gly
<400> 7
Phe Leu Asp Gly Ile Asp Lys Ala Gln Xaa Xaa His Xaa Lys Tyr His
1 5 10 15
Ser Asn Trp Arg Ala Met Ala Ser Asp Phe Asn Leu Pro Pro Val Val
20 25 30
Ala Lys Glu Ile Val Ala Ser Cys Asp Lys Cys Gln Leu Lys Gly Glu
35 40 45
Ala Met His Gly Gln Val Asp Cys Ser Pro Gly Ile Trp Gln Leu Xaa
50 55 60
Cys Thr His Leu Glu Gly Lys Val Ile Leu Val Ala Val His Val Ala
65 70 75 80
Ser Gly Tyr Ile Glu Ala Glu Val Ile Pro Ala Glu Thr Gly Gln Glu
85 90 95
Thr Ala Tyr Phe Leu Leu Lys Leu Ala Gly Arg Trp Pro Val Lys Thr
100 105 110
Ile His Thr Xaa Asn Gly Ser Asn Phe Thr Gly Ala Thr Val Arg Xaa
115 120 125
Ala Cys Trp Trp Ala Gly Ile Lys Gln Glu Phe Gly Ile Pro Tyr Asn
130 135 140
Pro Gln Ser Gln Gly Val Val Xaa Ser Met Asn Lys Glu Leu Lys Lys
145 150 155 160
Ile Ile Gly Gln Val Arg Asp Xaa Ala Xaa His Leu Lys Thr Ala Val
165 170 175
Gln Met Ala Val Phe Ile His Asn Phe Lys Arg Lys Gly Gly Ile Gly
180 185 190
Gly Tyr Ser Ala Gly Glu Arg Ile Val Asp Ile Ile Ala Thr Asp Ile
195 200 205
Gln Thr Lys Glu Leu Gln Lys Gln Ile Thr Lys Ile Gln Asn Phe Arg
210 215 220
Val Tyr Tyr Arg Asp Ser Arg Asn Pro Leu Trp Lys Gly Pro Ala Lys
225 230 235 240
Leu Leu Trp Lys Gly Glu Gly Ala Val Val Ile Gln Asp Asn Ser Asp
245 250 255
Ile Lys Val Val Pro Arg Arg Lys Ala Lys Ile Ile Arg Asp Tyr Gly
260 265 270
Lys Gln Met Ala Gly Asp Asp Cys Val Ala Ser Arg Gln Asp Glu Asp
275 280 285
<210> 8
<211> 288
<212> PRT
<213> Artificial Sequence
<220>
<223> Modified integrase with mutations in HIV-1 replication
<220>
<221> misc_feature
<222> (168)..(168)
<223> Xaa can be Gln, Leu, Ala
<400> 8
Phe Leu Asp Gly Ile Asp Lys Ala Gln Asp Glu His Glu Lys Tyr His
1 5 10 15
Ser Asn Trp Arg Ala Met Ala Ser Asp Phe Asn Leu Pro Pro Val Val
20 25 30
Ala Lys Glu Ile Val Ala Ser Cys Asp Lys Cys Gln Leu Lys Gly Glu
35 40 45
Ala Met His Gly Gln Val Asp Cys Ser Pro Gly Ile Trp Gln Leu Asp
50 55 60
Cys Thr His Leu Glu Gly Lys Val Ile Leu Val Ala Val His Val Ala
65 70 75 80
Ser Gly Tyr Ile Glu Ala Glu Val Ile Pro Ala Glu Thr Gly Gln Glu
85 90 95
Thr Ala Tyr Phe Leu Leu Lys Leu Ala Gly Arg Trp Pro Val Lys Thr
100 105 110
Ile His Thr Asp Asn Gly Ser Asn Phe Thr Gly Ala Thr Val Arg Ala
115 120 125
Ala Cys Trp Trp Ala Gly Ile Lys Gln Glu Phe Gly Ile Pro Tyr Asn
130 135 140
Pro Gln Ser Gln Gly Val Val Glu Ser Met Asn Lys Glu Leu Lys Lys
145 150 155 160
Ile Ile Gly Gln Val Arg Asp Xaa Ala Glu His Leu Lys Thr Ala Val
165 170 175
Gln Met Ala Val Phe Ile His Asn Phe Lys Arg Lys Gly Gly Ile Gly
180 185 190
Gly Tyr Ser Ala Gly Glu Arg Ile Val Asp Ile Ile Ala Thr Asp Ile
195 200 205
Gln Thr Lys Glu Leu Gln Lys Gln Ile Thr Lys Ile Gln Asn Phe Arg
210 215 220
Val Tyr Tyr Arg Asp Ser Arg Asn Pro Leu Trp Lys Gly Pro Ala Lys
225 230 235 240
Leu Leu Trp Lys Gly Glu Gly Ala Val Val Ile Gln Asp Asn Ser Asp
245 250 255
Ile Lys Val Val Pro Arg Arg Lys Ala Lys Ile Ile Arg Asp Tyr Gly
260 265 270
Lys Gln Met Ala Gly Asp Asp Cys Val Ala Ser Arg Gln Asp Glu Asp
275 280 285
<210> 9
<211> 594
<212> PRT
<213> Artificial Sequence
<220>
<223> Hyperactive PiggyBac aa sequence
<400> 9
Met Gly Ser Ser Leu Asp Asp Glu His Ile Leu Ser Ala Leu Leu Gln
1 5 10 15
Ser Asp Asp Glu Leu Val Gly Glu Asp Ser Asp Ser Glu Val Ser Asp
20 25 30
His Val Ser Glu Asp Asp Val Gln Ser Asp Thr Glu Glu Ala Phe Ile
35 40 45
Asp Glu Val His Glu Val Gln Pro Thr Ser Ser Gly Ser Glu Ile Leu
50 55 60
Asp Glu Gln Asn Val Ile Glu Gln Pro Gly Ser Ser Leu Ala Ser Asn
65 70 75 80
Arg Ile Leu Thr Leu Pro Gln Arg Thr Ile Arg Gly Lys Asn Lys His
85 90 95
Cys Trp Ser Thr Ser Lys Pro Thr Arg Arg Ser Arg Val Ser Ala Leu
100 105 110
Asn Ile Val Arg Ser Gln Arg Gly Pro Thr Arg Met Cys Arg Asn Ile
115 120 125
Tyr Asp Pro Leu Leu Cys Phe Lys Leu Phe Phe Thr Asp Glu Ile Ile
130 135 140
Ser Glu Ile Val Lys Trp Thr Asn Ala Glu Ile Ser Leu Lys Arg Arg
145 150 155 160
Glu Ser Met Thr Ser Ala Thr Phe Arg Asp Thr Asn Glu Asp Glu Ile
165 170 175
Tyr Ala Phe Phe Gly Ile Leu Val Met Thr Ala Val Arg Lys Asp Asn
180 185 190
His Met Ser Thr Asp Asp Leu Phe Asp Arg Ser Leu Ser Met Val Tyr
195 200 205
Val Ser Val Met Ser Arg Asp Arg Phe Asp Phe Leu Ile Arg Cys Leu
210 215 220
Arg Met Asp Asp Lys Ser Ile Arg Pro Thr Leu Arg Glu Asn Asp Val
225 230 235 240
Phe Thr Pro Val Arg Lys Ile Trp Asp Leu Phe Ile His Gln Cys Ile
245 250 255
Gln Asn Tyr Thr Pro Gly Ala His Leu Thr Ile Asp Glu Gln Leu Leu
260 265 270
Gly Phe Arg Gly Arg Cys Pro Phe Arg Val Tyr Ile Pro Asn Lys Pro
275 280 285
Ser Lys Tyr Gly Ile Lys Ile Leu Met Met Cys Asp Ser Gly Thr Lys
290 295 300
Tyr Met Ile Asn Gly Met Pro Tyr Leu Gly Arg Gly Thr Gln Thr Asn
305 310 315 320
Gly Val Pro Leu Gly Glu Tyr Tyr Val Lys Glu Leu Ser Lys Pro Val
325 330 335
His Gly Ser Cys Arg Asn Ile Thr Cys Asp Asn Trp Phe Thr Ser Ile
340 345 350
Pro Leu Ala Lys Asn Leu Leu Gln Glu Pro Tyr Lys Leu Thr Ile Val
355 360 365
Gly Thr Val Arg Ser Asn Lys Arg Glu Ile Pro Glu Val Leu Lys Asn
370 375 380
Ser Arg Ser Arg Pro Val Gly Thr Ser Met Phe Cys Phe Asp Gly Pro
385 390 395 400
Leu Thr Leu Val Ser Tyr Lys Pro Lys Pro Ala Lys Met Val Tyr Leu
405 410 415
Leu Ser Ser Cys Asp Glu Asp Ala Ser Ile Asn Glu Ser Thr Gly Lys
420 425 430
Pro Gln Met Val Met Tyr Tyr Asn Gln Thr Lys Gly Gly Val Asp Thr
435 440 445
Leu Asp Gln Met Cys Ser Val Met Thr Cys Ser Arg Lys Thr Asn Arg
450 455 460
Trp Pro Met Ala Leu Leu Tyr Gly Met Ile Asn Ile Ala Cys Ile Asn
465 470 475 480
Ser Phe Ile Ile Tyr Ser His Asn Val Ser Ser Lys Gly Glu Lys Val
485 490 495
Gln Ser Arg Lys Lys Phe Met Arg Asn Leu Tyr Met Gly Leu Thr Ser
500 505 510
Ser Phe Met Arg Lys Arg Leu Glu Ala Pro Thr Leu Lys Arg Tyr Leu
515 520 525
Arg Asp Asn Ile Ser Asn Ile Leu Pro Lys Glu Val Pro Gly Thr Ser
530 535 540
Asp Asp Ser Thr Glu Glu Pro Val Met Lys Lys Arg Thr Tyr Cys Thr
545 550 555 560
Tyr Cys Pro Ser Lys Ile Arg Arg Lys Ala Ser Ala Ser Cys Lys Lys
565 570 575
Cys Lys Lys Val Ile Cys Arg Glu His Asn Ile Asp Met Cys Gln Ser
580 585 590
Cys Phe
<210> 10
<211> 594
<212> PRT
<213> Artificial Sequence
<220>
<223> Modified hyperactive PiggyBac
<220>
<221> misc_feature
<222> (245)..(245)
<223> Wherein amino acid can be Arg, Ala
<220>
<221> misc_feature
<222> (268)..(268)
<223> Wherein amino acid can be Asp, Asn
<220>
<221> misc_feature
<222> (275)..(275)
<223> Wherein amino acid can be Arg, Ala
<220>
<221> misc_feature
<222> (277)..(277)
<223> Wherein amino acid can be Arg, Ala
<220>
<221> misc_feature
<222> (287)..(287)
<223> Wherein amino acid can be Ala, Lys
<220>
<221> misc_feature
<222> (290)..(290)
<223> Wherein amino acid can be Ala, Lys
<220>
<221> misc_feature
<222> (315)..(315)
<223> Wherein amino acid can be Arg, Ala
<220>
<221> misc_feature
<222> (325)..(325)
<223> Wherein amino acid can be Gly, Ala
<220>
<221> misc_feature
<222> (341)..(341)
<223> Wherein amino acid can be Arg, Ala
<220>
<221> misc_feature
<222> (346)..(346)
<223> Wherein amino acid can be Asp, Asn
<220>
<221> misc_feature
<222> (347)..(347)
<223> Wherein amino acid can be Asn, Ala, Ser
<220>
<221> misc_feature
<222> (350)..(350)
<223> Wherein amino acid can be Thr, Ala
<220>
<221> misc_feature
<222> (351)..(351)
<223> Xaa can be Ser, Glu, Pro, Ala
<220>
<221> misc_feature
<222> (356)..(356)
<223> Wherein amino acid can be Lys, Glu
<220>
<221> misc_feature
<222> (357)..(357)
<223> Wherein amino acid can be Asn, Ala
<220>
<221> misc_feature
<222> (372)..(372)
<223> Wherein amino acid can be Arg, Ala
<220>
<221> misc_feature
<222> (375)..(375)
<223> Wherein amino acid can be Lys, Ala
<220>
<221> misc_feature
<222> (388)..(388)
<223> Wherein amino acid can be Arg, Ala
<220>
<221> misc_feature
<222> (409)..(409)
<223> Wherein amino acid can be Lys, Ala
<220>
<221> misc_feature
<222> (412)..(412)
<223> Wherein amino acid can be Lys, Ala
<220>
<221> misc_feature
<222> (432)..(432)
<223> Wherein amino acid can be Lys, Ala
<220>
<221> misc_feature
<222> (460)..(460)
<223> Wherein amino acid can be Arg, Ala
<220>
<221> misc_feature
<222> (461)..(461)
<223> Wherein amino acid can be Ala, Lys
<220>
<221> misc_feature
<222> (465)..(465)
<223> Wherein amino acid can be Trp, Ala
<220>
<221> misc_feature
<222> (560)..(560)
<223> Wherein amino acid can be Thr, Ala
<220>
<221> misc_feature
<222> (564)..(564)
<223> Wherein amino acid can be Ser, Pro
<220>
<221> misc_feature
<222> (571)..(571)
<223> Wherein amino acid can be Asn, Ser
<220>
<221> misc_feature
<222> (573)..(573)
<223> Wherein amino acid can be Ser, Ala
<220>
<221> misc_feature
<222> (576)..(576)
<223> Wherein amino acid can be Lys, Ala
<220>
<221> misc_feature
<222> (586)..(586)
<223> Wherein amino acid can be His, any naturally occuring amino acid
<220>
<221> misc_feature
<222> (587)..(587)
<223> Wherein amino acid can be Ile, Ala
<220>
<221> misc_feature
<222> (589)..(589)
<223> Wherein amino acid can be Met, Val
<220>
<221> misc_feature
<222> (592)..(592)
<223> Wherein amino acid can be Ser, Gly
<220>
<221> misc_feature
<222> (594)..(594)
<223> Wherein amino acid can be Phe, Leu
<400> 10
Met Gly Ser Ser Leu Asp Asp Glu His Ile Leu Ser Ala Leu Leu Gln
1 5 10 15
Ser Asp Asp Glu Leu Val Gly Glu Asp Ser Asp Ser Glu Val Ser Asp
20 25 30
His Val Ser Glu Asp Asp Val Gln Ser Asp Thr Glu Glu Ala Phe Ile
35 40 45
Asp Glu Val His Glu Val Gln Pro Thr Ser Ser Gly Ser Glu Ile Leu
50 55 60
Asp Glu Gln Asn Val Ile Glu Gln Pro Gly Ser Ser Leu Ala Ser Asn
65 70 75 80
Arg Ile Leu Thr Leu Pro Gln Arg Thr Ile Arg Gly Lys Asn Lys His
85 90 95
Cys Trp Ser Thr Ser Lys Pro Thr Arg Arg Ser Arg Val Ser Ala Leu
100 105 110
Asn Ile Val Arg Ser Gln Arg Gly Pro Thr Arg Met Cys Arg Asn Ile
115 120 125
Tyr Asp Pro Leu Leu Cys Phe Lys Leu Phe Phe Thr Asp Glu Ile Ile
130 135 140
Ser Glu Ile Val Lys Trp Thr Asn Ala Glu Ile Ser Leu Lys Arg Arg
145 150 155 160
Glu Ser Met Thr Ser Ala Thr Phe Arg Asp Thr Asn Glu Asp Glu Ile
165 170 175
Tyr Ala Phe Phe Gly Ile Leu Val Met Thr Ala Val Arg Lys Asp Asn
180 185 190
His Met Ser Thr Asp Asp Leu Phe Asp Arg Ser Leu Ser Met Val Tyr
195 200 205
Val Ser Val Met Ser Arg Asp Arg Phe Asp Phe Leu Ile Arg Cys Leu
210 215 220
Arg Met Asp Asp Lys Ser Ile Arg Pro Thr Leu Arg Glu Asn Asp Val
225 230 235 240
Phe Thr Pro Val Arg Lys Ile Trp Asp Leu Phe Ile His Gln Cys Ile
245 250 255
Gln Asn Tyr Thr Pro Gly Ala His Leu Thr Ile Asp Glu Gln Leu Leu
260 265 270
Gly Phe Arg Gly Arg Cys Pro Phe Arg Val Tyr Ile Pro Asn Lys Pro
275 280 285
Ser Lys Tyr Gly Ile Lys Ile Leu Met Met Cys Asp Ser Gly Thr Lys
290 295 300
Tyr Met Ile Asn Gly Met Pro Tyr Leu Gly Arg Gly Thr Gln Thr Asn
305 310 315 320
Gly Val Pro Leu Gly Glu Tyr Tyr Val Lys Glu Leu Ser Lys Pro Val
325 330 335
His Gly Ser Cys Arg Asn Ile Thr Cys Asp Asn Trp Phe Thr Ser Ile
340 345 350
Pro Leu Ala Lys Asn Leu Leu Gln Glu Pro Tyr Lys Leu Thr Ile Val
355 360 365
Gly Thr Val Arg Ser Asn Lys Arg Glu Ile Pro Glu Val Leu Lys Asn
370 375 380
Ser Arg Ser Arg Pro Val Gly Thr Ser Met Phe Cys Phe Asp Gly Pro
385 390 395 400
Leu Thr Leu Val Ser Tyr Lys Pro Lys Pro Ala Lys Met Val Tyr Leu
405 410 415
Leu Ser Ser Cys Asp Glu Asp Ala Ser Ile Asn Glu Ser Thr Gly Lys
420 425 430
Pro Gln Met Val Met Tyr Tyr Asn Gln Thr Lys Gly Gly Val Asp Thr
435 440 445
Leu Asp Gln Met Cys Ser Val Met Thr Cys Ser Arg Lys Thr Asn Arg
450 455 460
Trp Pro Met Ala Leu Leu Tyr Gly Met Ile Asn Ile Ala Cys Ile Asn
465 470 475 480
Ser Phe Ile Ile Tyr Ser His Asn Val Ser Ser Lys Gly Glu Lys Val
485 490 495
Gln Ser Arg Lys Lys Phe Met Arg Asn Leu Tyr Met Gly Leu Thr Ser
500 505 510
Ser Phe Met Arg Lys Arg Leu Glu Ala Pro Thr Leu Lys Arg Tyr Leu
515 520 525
Arg Asp Asn Ile Ser Asn Ile Leu Pro Lys Glu Val Pro Gly Thr Ser
530 535 540
Asp Asp Ser Thr Glu Glu Pro Val Met Lys Lys Arg Thr Tyr Cys Thr
545 550 555 560
Tyr Cys Pro Ser Lys Ile Arg Arg Lys Ala Ser Ala Ser Cys Lys Lys
565 570 575
Cys Lys Lys Val Ile Cys Arg Glu His Asn Ile Asp Met Cys Gln Ser
580 585 590
Cys Phe
<210> 11
<211> 594
<212> PRT
<213> Artificial Sequence
<220>
<223> Modified hyperactive PiggyBac with mutations in the catalytic
triad
<220>
<221> misc_feature
<222> (268)..(268)
<223> Xaa can be Asp, Asn
<220>
<221> misc_feature
<222> (346)..(346)
<223> Xaa can be Asp, Asn
<400> 11
Met Gly Ser Ser Leu Asp Asp Glu His Ile Leu Ser Ala Leu Leu Gln
1 5 10 15
Ser Asp Asp Glu Leu Val Gly Glu Asp Ser Asp Ser Glu Val Ser Asp
20 25 30
His Val Ser Glu Asp Asp Val Gln Ser Asp Thr Glu Glu Ala Phe Ile
35 40 45
Asp Glu Val His Glu Val Gln Pro Thr Ser Ser Gly Ser Glu Ile Leu
50 55 60
Asp Glu Gln Asn Val Ile Glu Gln Pro Gly Ser Ser Leu Ala Ser Asn
65 70 75 80
Arg Ile Leu Thr Leu Pro Gln Arg Thr Ile Arg Gly Lys Asn Lys His
85 90 95
Cys Trp Ser Thr Ser Lys Pro Thr Arg Arg Ser Arg Val Ser Ala Leu
100 105 110
Asn Ile Val Arg Ser Gln Arg Gly Pro Thr Arg Met Cys Arg Asn Ile
115 120 125
Tyr Asp Pro Leu Leu Cys Phe Lys Leu Phe Phe Thr Asp Glu Ile Ile
130 135 140
Ser Glu Ile Val Lys Trp Thr Asn Ala Glu Ile Ser Leu Lys Arg Arg
145 150 155 160
Glu Ser Met Thr Ser Ala Thr Phe Arg Asp Thr Asn Glu Asp Glu Ile
165 170 175
Tyr Ala Phe Phe Gly Ile Leu Val Met Thr Ala Val Arg Lys Asp Asn
180 185 190
His Met Ser Thr Asp Asp Leu Phe Asp Arg Ser Leu Ser Met Val Tyr
195 200 205
Val Ser Val Met Ser Arg Asp Arg Phe Asp Phe Leu Ile Arg Cys Leu
210 215 220
Arg Met Asp Asp Lys Ser Ile Arg Pro Thr Leu Arg Glu Asn Asp Val
225 230 235 240
Phe Thr Pro Val Arg Lys Ile Trp Asp Leu Phe Ile His Gln Cys Ile
245 250 255
Gln Asn Tyr Thr Pro Gly Ala His Leu Thr Ile Xaa Glu Gln Leu Leu
260 265 270
Gly Phe Arg Gly Arg Cys Pro Phe Arg Val Tyr Ile Pro Asn Lys Pro
275 280 285
Ser Lys Tyr Gly Ile Lys Ile Leu Met Met Cys Asp Ser Gly Thr Lys
290 295 300
Tyr Met Ile Asn Gly Met Pro Tyr Leu Gly Arg Gly Thr Gln Thr Asn
305 310 315 320
Gly Val Pro Leu Gly Glu Tyr Tyr Val Lys Glu Leu Ser Lys Pro Val
325 330 335
His Gly Ser Cys Arg Asn Ile Thr Cys Xaa Asn Trp Phe Thr Ser Ile
340 345 350
Pro Leu Ala Lys Asn Leu Leu Gln Glu Pro Tyr Lys Leu Thr Ile Val
355 360 365
Gly Thr Val Arg Ser Asn Lys Arg Glu Ile Pro Glu Val Leu Lys Asn
370 375 380
Ser Arg Ser Arg Pro Val Gly Thr Ser Met Phe Cys Phe Asp Gly Pro
385 390 395 400
Leu Thr Leu Val Ser Tyr Lys Pro Lys Pro Ala Lys Met Val Tyr Leu
405 410 415
Leu Ser Ser Cys Asp Glu Asp Ala Ser Ile Asn Glu Ser Thr Gly Lys
420 425 430
Pro Gln Met Val Met Tyr Tyr Asn Gln Thr Lys Gly Gly Val Asp Thr
435 440 445
Leu Asp Gln Met Cys Ser Val Met Thr Cys Ser Arg Lys Thr Asn Arg
450 455 460
Trp Pro Met Ala Leu Leu Tyr Gly Met Ile Asn Ile Ala Cys Ile Asn
465 470 475 480
Ser Phe Ile Ile Tyr Ser His Asn Val Ser Ser Lys Gly Glu Lys Val
485 490 495
Gln Ser Arg Lys Lys Phe Met Arg Asn Leu Tyr Met Gly Leu Thr Ser
500 505 510
Ser Phe Met Arg Lys Arg Leu Glu Ala Pro Thr Leu Lys Arg Tyr Leu
515 520 525
Arg Asp Asn Ile Ser Asn Ile Leu Pro Lys Glu Val Pro Gly Thr Ser
530 535 540
Asp Asp Ser Thr Glu Glu Pro Val Met Lys Lys Arg Thr Tyr Cys Thr
545 550 555 560
Tyr Cys Pro Ser Lys Ile Arg Arg Lys Ala Ser Ala Ser Cys Lys Lys
565 570 575
Cys Lys Lys Val Ile Cys Arg Glu His Asn Ile Asp Met Cys Gln Ser
580 585 590
Cys Phe
<210> 12
<211> 594
<212> PRT
<213> Artificial Sequence
<220>
<223> Modified hyperactive PiggyBac with mutations in amino acids that
are critical for excision
<220>
<221> misc_feature
<222> (287)..(287)
<223> Xaa can be Lys, Ala
<220>
<221> misc_feature
<222> (290)..(290)
<223> Xaa can be Lys, Ala
<220>
<221> misc_feature
<222> (460)..(460)
<223> Xaa can be Arg, Ala
<220>
<221> misc_feature
<222> (461)..(461)
<223> Xaa can be Lys, Ala
<400> 12
Met Gly Ser Ser Leu Asp Asp Glu His Ile Leu Ser Ala Leu Leu Gln
1 5 10 15
Ser Asp Asp Glu Leu Val Gly Glu Asp Ser Asp Ser Glu Val Ser Asp
20 25 30
His Val Ser Glu Asp Asp Val Gln Ser Asp Thr Glu Glu Ala Phe Ile
35 40 45
Asp Glu Val His Glu Val Gln Pro Thr Ser Ser Gly Ser Glu Ile Leu
50 55 60
Asp Glu Gln Asn Val Ile Glu Gln Pro Gly Ser Ser Leu Ala Ser Asn
65 70 75 80
Arg Ile Leu Thr Leu Pro Gln Arg Thr Ile Arg Gly Lys Asn Lys His
85 90 95
Cys Trp Ser Thr Ser Lys Pro Thr Arg Arg Ser Arg Val Ser Ala Leu
100 105 110
Asn Ile Val Arg Ser Gln Arg Gly Pro Thr Arg Met Cys Arg Asn Ile
115 120 125
Tyr Asp Pro Leu Leu Cys Phe Lys Leu Phe Phe Thr Asp Glu Ile Ile
130 135 140
Ser Glu Ile Val Lys Trp Thr Asn Ala Glu Ile Ser Leu Lys Arg Arg
145 150 155 160
Glu Ser Met Thr Ser Ala Thr Phe Arg Asp Thr Asn Glu Asp Glu Ile
165 170 175
Tyr Ala Phe Phe Gly Ile Leu Val Met Thr Ala Val Arg Lys Asp Asn
180 185 190
His Met Ser Thr Asp Asp Leu Phe Asp Arg Ser Leu Ser Met Val Tyr
195 200 205
Val Ser Val Met Ser Arg Asp Arg Phe Asp Phe Leu Ile Arg Cys Leu
210 215 220
Arg Met Asp Asp Lys Ser Ile Arg Pro Thr Leu Arg Glu Asn Asp Val
225 230 235 240
Phe Thr Pro Val Arg Lys Ile Trp Asp Leu Phe Ile His Gln Cys Ile
245 250 255
Gln Asn Tyr Thr Pro Gly Ala His Leu Thr Ile Asp Glu Gln Leu Leu
260 265 270
Gly Phe Arg Gly Arg Cys Pro Phe Arg Val Tyr Ile Pro Asn Xaa Pro
275 280 285
Ser Xaa Tyr Gly Ile Lys Ile Leu Met Met Cys Asp Ser Gly Thr Lys
290 295 300
Tyr Met Ile Asn Gly Met Pro Tyr Leu Gly Arg Gly Thr Gln Thr Asn
305 310 315 320
Gly Val Pro Leu Gly Glu Tyr Tyr Val Lys Glu Leu Ser Lys Pro Val
325 330 335
His Gly Ser Cys Arg Asn Ile Thr Cys Asp Asn Trp Phe Thr Ser Ile
340 345 350
Pro Leu Ala Lys Asn Leu Leu Gln Glu Pro Tyr Lys Leu Thr Ile Val
355 360 365
Gly Thr Val Arg Ser Asn Lys Arg Glu Ile Pro Glu Val Leu Lys Asn
370 375 380
Ser Arg Ser Arg Pro Val Gly Thr Ser Met Phe Cys Phe Asp Gly Pro
385 390 395 400
Leu Thr Leu Val Ser Tyr Lys Pro Lys Pro Ala Lys Met Val Tyr Leu
405 410 415
Leu Ser Ser Cys Asp Glu Asp Ala Ser Ile Asn Glu Ser Thr Gly Lys
420 425 430
Pro Gln Met Val Met Tyr Tyr Asn Gln Thr Lys Gly Gly Val Asp Thr
435 440 445
Leu Asp Gln Met Cys Ser Val Met Thr Cys Ser Xaa Xaa Thr Asn Arg
450 455 460
Trp Pro Met Ala Leu Leu Tyr Gly Met Ile Asn Ile Ala Cys Ile Asn
465 470 475 480
Ser Phe Ile Ile Tyr Ser His Asn Val Ser Ser Lys Gly Glu Lys Val
485 490 495
Gln Ser Arg Lys Lys Phe Met Arg Asn Leu Tyr Met Gly Leu Thr Ser
500 505 510
Ser Phe Met Arg Lys Arg Leu Glu Ala Pro Thr Leu Lys Arg Tyr Leu
515 520 525
Arg Asp Asn Ile Ser Asn Ile Leu Pro Lys Glu Val Pro Gly Thr Ser
530 535 540
Asp Asp Ser Thr Glu Glu Pro Val Met Lys Lys Arg Thr Tyr Cys Thr
545 550 555 560
Tyr Cys Pro Ser Lys Ile Arg Arg Lys Ala Ser Ala Ser Cys Lys Lys
565 570 575
Cys Lys Lys Val Ile Cys Arg Glu His Asn Ile Asp Met Cys Gln Ser
580 585 590
Cys Phe
<210> 13
<211> 594
<212> PRT
<213> Artificial Sequence
<220>
<223> Modified hyperactive PiggyBac with mutation that are involved in
target joining
<220>
<221> misc_feature
<222> (351)..(351)
<223> Xaa can be Ser, Glu, Pro, Ala
<220>
<221> misc_feature
<222> (356)..(356)
<223> Xaa can be Lys, Glu
<400> 13
Met Gly Ser Ser Leu Asp Asp Glu His Ile Leu Ser Ala Leu Leu Gln
1 5 10 15
Ser Asp Asp Glu Leu Val Gly Glu Asp Ser Asp Ser Glu Val Ser Asp
20 25 30
His Val Ser Glu Asp Asp Val Gln Ser Asp Thr Glu Glu Ala Phe Ile
35 40 45
Asp Glu Val His Glu Val Gln Pro Thr Ser Ser Gly Ser Glu Ile Leu
50 55 60
Asp Glu Gln Asn Val Ile Glu Gln Pro Gly Ser Ser Leu Ala Ser Asn
65 70 75 80
Arg Ile Leu Thr Leu Pro Gln Arg Thr Ile Arg Gly Lys Asn Lys His
85 90 95
Cys Trp Ser Thr Ser Lys Pro Thr Arg Arg Ser Arg Val Ser Ala Leu
100 105 110
Asn Ile Val Arg Ser Gln Arg Gly Pro Thr Arg Met Cys Arg Asn Ile
115 120 125
Tyr Asp Pro Leu Leu Cys Phe Lys Leu Phe Phe Thr Asp Glu Ile Ile
130 135 140
Ser Glu Ile Val Lys Trp Thr Asn Ala Glu Ile Ser Leu Lys Arg Arg
145 150 155 160
Glu Ser Met Thr Ser Ala Thr Phe Arg Asp Thr Asn Glu Asp Glu Ile
165 170 175
Tyr Ala Phe Phe Gly Ile Leu Val Met Thr Ala Val Arg Lys Asp Asn
180 185 190
His Met Ser Thr Asp Asp Leu Phe Asp Arg Ser Leu Ser Met Val Tyr
195 200 205
Val Ser Val Met Ser Arg Asp Arg Phe Asp Phe Leu Ile Arg Cys Leu
210 215 220
Arg Met Asp Asp Lys Ser Ile Arg Pro Thr Leu Arg Glu Asn Asp Val
225 230 235 240
Phe Thr Pro Val Arg Lys Ile Trp Asp Leu Phe Ile His Gln Cys Ile
245 250 255
Gln Asn Tyr Thr Pro Gly Ala His Leu Thr Ile Asp Glu Gln Leu Leu
260 265 270
Gly Phe Arg Gly Arg Cys Pro Phe Arg Val Tyr Ile Pro Asn Lys Pro
275 280 285
Ser Lys Tyr Gly Ile Lys Ile Leu Met Met Cys Asp Ser Gly Thr Lys
290 295 300
Tyr Met Ile Asn Gly Met Pro Tyr Leu Gly Arg Gly Thr Gln Thr Asn
305 310 315 320
Gly Val Pro Leu Gly Glu Tyr Tyr Val Lys Glu Leu Ser Lys Pro Val
325 330 335
His Gly Ser Cys Arg Asn Ile Thr Cys Asp Asn Trp Phe Thr Xaa Ile
340 345 350
Pro Leu Ala Xaa Asn Leu Leu Gln Glu Pro Tyr Lys Leu Thr Ile Val
355 360 365
Gly Thr Val Arg Ser Asn Lys Arg Glu Ile Pro Glu Val Leu Lys Asn
370 375 380
Ser Arg Ser Arg Pro Val Gly Thr Ser Met Phe Cys Phe Asp Gly Pro
385 390 395 400
Leu Thr Leu Val Ser Tyr Lys Pro Lys Pro Ala Lys Met Val Tyr Leu
405 410 415
Leu Ser Ser Cys Asp Glu Asp Ala Ser Ile Asn Glu Ser Thr Gly Lys
420 425 430
Pro Gln Met Val Met Tyr Tyr Asn Gln Thr Lys Gly Gly Val Asp Thr
435 440 445
Leu Asp Gln Met Cys Ser Val Met Thr Cys Ser Arg Lys Thr Asn Arg
450 455 460
Trp Pro Met Ala Leu Leu Tyr Gly Met Ile Asn Ile Ala Cys Ile Asn
465 470 475 480
Ser Phe Ile Ile Tyr Ser His Asn Val Ser Ser Lys Gly Glu Lys Val
485 490 495
Gln Ser Arg Lys Lys Phe Met Arg Asn Leu Tyr Met Gly Leu Thr Ser
500 505 510
Ser Phe Met Arg Lys Arg Leu Glu Ala Pro Thr Leu Lys Arg Tyr Leu
515 520 525
Arg Asp Asn Ile Ser Asn Ile Leu Pro Lys Glu Val Pro Gly Thr Ser
530 535 540
Asp Asp Ser Thr Glu Glu Pro Val Met Lys Lys Arg Thr Tyr Cys Thr
545 550 555 560
Tyr Cys Pro Ser Lys Ile Arg Arg Lys Ala Ser Ala Ser Cys Lys Lys
565 570 575
Cys Lys Lys Val Ile Cys Arg Glu His Asn Ile Asp Met Cys Gln Ser
580 585 590
Cys Phe
<210> 14
<211> 594
<212> PRT
<213> Artificial Sequence
<220>
<223> Modified hyperactive PiggyBac with mutations that are critical
for integration
<220>
<221> misc_feature
<222> (560)..(560)
<223> Wherein amino acid can be Thr, Ala,
<220>
<221> misc_feature
<222> (564)..(564)
<223> Wherein amino acid can be Ser, Pro
<220>
<221> misc_feature
<222> (571)..(571)
<223> Wherein amino acid can be Asn, Ser
<220>
<221> misc_feature
<222> (573)..(573)
<223> Wherein amino acid can be Ser, Ala
<220>
<221> misc_feature
<222> (589)..(589)
<223> Wherein amino acid can be Met, Val
<220>
<221> misc_feature
<222> (592)..(592)
<223> Wherein amino acid can be Ser, Gly
<220>
<221> misc_feature
<222> (594)..(594)
<223> Wherein amino acid can be Phe, Leu
<400> 14
Met Gly Ser Ser Leu Asp Asp Glu His Ile Leu Ser Ala Leu Leu Gln
1 5 10 15
Ser Asp Asp Glu Leu Val Gly Glu Asp Ser Asp Ser Glu Val Ser Asp
20 25 30
His Val Ser Glu Asp Asp Val Gln Ser Asp Thr Glu Glu Ala Phe Ile
35 40 45
Asp Glu Val His Glu Val Gln Pro Thr Ser Ser Gly Ser Glu Ile Leu
50 55 60
Asp Glu Gln Asn Val Ile Glu Gln Pro Gly Ser Ser Leu Ala Ser Asn
65 70 75 80
Arg Ile Leu Thr Leu Pro Gln Arg Thr Ile Arg Gly Lys Asn Lys His
85 90 95
Cys Trp Ser Thr Ser Lys Pro Thr Arg Arg Ser Arg Val Ser Ala Leu
100 105 110
Asn Ile Val Arg Ser Gln Arg Gly Pro Thr Arg Met Cys Arg Asn Ile
115 120 125
Tyr Asp Pro Leu Leu Cys Phe Lys Leu Phe Phe Thr Asp Glu Ile Ile
130 135 140
Ser Glu Ile Val Lys Trp Thr Asn Ala Glu Ile Ser Leu Lys Arg Arg
145 150 155 160
Glu Ser Met Thr Ser Ala Thr Phe Arg Asp Thr Asn Glu Asp Glu Ile
165 170 175
Tyr Ala Phe Phe Gly Ile Leu Val Met Thr Ala Val Arg Lys Asp Asn
180 185 190
His Met Ser Thr Asp Asp Leu Phe Asp Arg Ser Leu Ser Met Val Tyr
195 200 205
Val Ser Val Met Ser Arg Asp Arg Phe Asp Phe Leu Ile Arg Cys Leu
210 215 220
Arg Met Asp Asp Lys Ser Ile Arg Pro Thr Leu Arg Glu Asn Asp Val
225 230 235 240
Phe Thr Pro Val Arg Lys Ile Trp Asp Leu Phe Ile His Gln Cys Ile
245 250 255
Gln Asn Tyr Thr Pro Gly Ala His Leu Thr Ile Asp Glu Gln Leu Leu
260 265 270
Gly Phe Arg Gly Arg Cys Pro Phe Arg Val Tyr Ile Pro Asn Lys Pro
275 280 285
Ser Lys Tyr Gly Ile Lys Ile Leu Met Met Cys Asp Ser Gly Thr Lys
290 295 300
Tyr Met Ile Asn Gly Met Pro Tyr Leu Gly Arg Gly Thr Gln Thr Asn
305 310 315 320
Gly Val Pro Leu Gly Glu Tyr Tyr Val Lys Glu Leu Ser Lys Pro Val
325 330 335
His Gly Ser Cys Arg Asn Ile Thr Cys Asp Asn Trp Phe Thr Ser Ile
340 345 350
Pro Leu Ala Lys Asn Leu Leu Gln Glu Pro Tyr Lys Leu Thr Ile Val
355 360 365
Gly Thr Val Arg Ser Asn Lys Arg Glu Ile Pro Glu Val Leu Lys Asn
370 375 380
Ser Arg Ser Arg Pro Val Gly Thr Ser Met Phe Cys Phe Asp Gly Pro
385 390 395 400
Leu Thr Leu Val Ser Tyr Lys Pro Lys Pro Ala Lys Met Val Tyr Leu
405 410 415
Leu Ser Ser Cys Asp Glu Asp Ala Ser Ile Asn Glu Ser Thr Gly Lys
420 425 430
Pro Gln Met Val Met Tyr Tyr Asn Gln Thr Lys Gly Gly Val Asp Thr
435 440 445
Leu Asp Gln Met Cys Ser Val Met Thr Cys Ser Arg Lys Thr Asn Arg
450 455 460
Trp Pro Met Ala Leu Leu Tyr Gly Met Ile Asn Ile Ala Cys Ile Asn
465 470 475 480
Ser Phe Ile Ile Tyr Ser His Asn Val Ser Ser Lys Gly Glu Lys Val
485 490 495
Gln Ser Arg Lys Lys Phe Met Arg Asn Leu Tyr Met Gly Leu Thr Ser
500 505 510
Ser Phe Met Arg Lys Arg Leu Glu Ala Pro Thr Leu Lys Arg Tyr Leu
515 520 525
Arg Asp Asn Ile Ser Asn Ile Leu Pro Lys Glu Val Pro Gly Thr Ser
530 535 540
Asp Asp Ser Thr Glu Glu Pro Val Met Lys Lys Arg Thr Tyr Cys Thr
545 550 555 560
Tyr Cys Pro Ser Lys Ile Arg Arg Lys Ala Ser Ala Ser Cys Lys Lys
565 570 575
Cys Lys Lys Val Ile Cys Arg Glu His Asn Ile Asp Met Cys Gln Ser
580 585 590
Cys Phe
<210> 15
<211> 594
<212> PRT
<213> Artificial Sequence
<220>
<223> Modified hyperactive PiggyBac with mutations that are involved in
alignment
<220>
<221> misc_feature
<222> (325)..(325)
<223> Xaa can be Gly, Ala
<220>
<221> misc_feature
<222> (347)..(347)
<223> Xaa can be Asn, Ala, Ser
<220>
<221> misc_feature
<222> (350)..(350)
<223> Xaa can be Thr, Ala
<220>
<221> misc_feature
<222> (465)..(465)
<223> Xaa can be Trp, Ala
<400> 15
Met Gly Ser Ser Leu Asp Asp Glu His Ile Leu Ser Ala Leu Leu Gln
1 5 10 15
Ser Asp Asp Glu Leu Val Gly Glu Asp Ser Asp Ser Glu Val Ser Asp
20 25 30
His Val Ser Glu Asp Asp Val Gln Ser Asp Thr Glu Glu Ala Phe Ile
35 40 45
Asp Glu Val His Glu Val Gln Pro Thr Ser Ser Gly Ser Glu Ile Leu
50 55 60
Asp Glu Gln Asn Val Ile Glu Gln Pro Gly Ser Ser Leu Ala Ser Asn
65 70 75 80
Arg Ile Leu Thr Leu Pro Gln Arg Thr Ile Arg Gly Lys Asn Lys His
85 90 95
Cys Trp Ser Thr Ser Lys Pro Thr Arg Arg Ser Arg Val Ser Ala Leu
100 105 110
Asn Ile Val Arg Ser Gln Arg Gly Pro Thr Arg Met Cys Arg Asn Ile
115 120 125
Tyr Asp Pro Leu Leu Cys Phe Lys Leu Phe Phe Thr Asp Glu Ile Ile
130 135 140
Ser Glu Ile Val Lys Trp Thr Asn Ala Glu Ile Ser Leu Lys Arg Arg
145 150 155 160
Glu Ser Met Thr Ser Ala Thr Phe Arg Asp Thr Asn Glu Asp Glu Ile
165 170 175
Tyr Ala Phe Phe Gly Ile Leu Val Met Thr Ala Val Arg Lys Asp Asn
180 185 190
His Met Ser Thr Asp Asp Leu Phe Asp Arg Ser Leu Ser Met Val Tyr
195 200 205
Val Ser Val Met Ser Arg Asp Arg Phe Asp Phe Leu Ile Arg Cys Leu
210 215 220
Arg Met Asp Asp Lys Ser Ile Arg Pro Thr Leu Arg Glu Asn Asp Val
225 230 235 240
Phe Thr Pro Val Arg Lys Ile Trp Asp Leu Phe Ile His Gln Cys Ile
245 250 255
Gln Asn Tyr Thr Pro Gly Ala His Leu Thr Ile Asp Glu Gln Leu Leu
260 265 270
Gly Phe Arg Gly Arg Cys Pro Phe Arg Val Tyr Ile Pro Asn Lys Pro
275 280 285
Ser Lys Tyr Gly Ile Lys Ile Leu Met Met Cys Asp Ser Gly Thr Lys
290 295 300
Tyr Met Ile Asn Gly Met Pro Tyr Leu Gly Arg Gly Thr Gln Thr Asn
305 310 315 320
Gly Val Pro Leu Xaa Glu Tyr Tyr Val Lys Glu Leu Ser Lys Pro Val
325 330 335
His Gly Ser Cys Arg Asn Ile Thr Cys Asp Xaa Trp Phe Xaa Ser Ile
340 345 350
Pro Leu Ala Lys Asn Leu Leu Gln Glu Pro Tyr Lys Leu Thr Ile Val
355 360 365
Gly Thr Val Arg Ser Asn Lys Arg Glu Ile Pro Glu Val Leu Lys Asn
370 375 380
Ser Arg Ser Arg Pro Val Gly Thr Ser Met Phe Cys Phe Asp Gly Pro
385 390 395 400
Leu Thr Leu Val Ser Tyr Lys Pro Lys Pro Ala Lys Met Val Tyr Leu
405 410 415
Leu Ser Ser Cys Asp Glu Asp Ala Ser Ile Asn Glu Ser Thr Gly Lys
420 425 430
Pro Gln Met Val Met Tyr Tyr Asn Gln Thr Lys Gly Gly Val Asp Thr
435 440 445
Leu Asp Gln Met Cys Ser Val Met Thr Cys Ser Arg Lys Thr Asn Arg
450 455 460
Xaa Pro Met Ala Leu Leu Tyr Gly Met Ile Asn Ile Ala Cys Ile Asn
465 470 475 480
Ser Phe Ile Ile Tyr Ser His Asn Val Ser Ser Lys Gly Glu Lys Val
485 490 495
Gln Ser Arg Lys Lys Phe Met Arg Asn Leu Tyr Met Gly Leu Thr Ser
500 505 510
Ser Phe Met Arg Lys Arg Leu Glu Ala Pro Thr Leu Lys Arg Tyr Leu
515 520 525
Arg Asp Asn Ile Ser Asn Ile Leu Pro Lys Glu Val Pro Gly Thr Ser
530 535 540
Asp Asp Ser Thr Glu Glu Pro Val Met Lys Lys Arg Thr Tyr Cys Thr
545 550 555 560
Tyr Cys Pro Ser Lys Ile Arg Arg Lys Ala Ser Ala Ser Cys Lys Lys
565 570 575
Cys Lys Lys Val Ile Cys Arg Glu His Asn Ile Asp Met Cys Gln Ser
580 585 590
Cys Phe
<210> 16
<211> 594
<212> PRT
<213> Artificial Sequence
<220>
<223> Modified hyperactive PiggyBac with mutations at well conserved
amino acids
<220>
<221> misc_feature
<222> (576)..(576)
<223> Xaa can be lys, Ala
<220>
<221> misc_feature
<222> (587)..(587)
<223> Xaa can be Ile, Ala
<400> 16
Met Gly Ser Ser Leu Asp Asp Glu His Ile Leu Ser Ala Leu Leu Gln
1 5 10 15
Ser Asp Asp Glu Leu Val Gly Glu Asp Ser Asp Ser Glu Val Ser Asp
20 25 30
His Val Ser Glu Asp Asp Val Gln Ser Asp Thr Glu Glu Ala Phe Ile
35 40 45
Asp Glu Val His Glu Val Gln Pro Thr Ser Ser Gly Ser Glu Ile Leu
50 55 60
Asp Glu Gln Asn Val Ile Glu Gln Pro Gly Ser Ser Leu Ala Ser Asn
65 70 75 80
Arg Ile Leu Thr Leu Pro Gln Arg Thr Ile Arg Gly Lys Asn Lys His
85 90 95
Cys Trp Ser Thr Ser Lys Pro Thr Arg Arg Ser Arg Val Ser Ala Leu
100 105 110
Asn Ile Val Arg Ser Gln Arg Gly Pro Thr Arg Met Cys Arg Asn Ile
115 120 125
Tyr Asp Pro Leu Leu Cys Phe Lys Leu Phe Phe Thr Asp Glu Ile Ile
130 135 140
Ser Glu Ile Val Lys Trp Thr Asn Ala Glu Ile Ser Leu Lys Arg Arg
145 150 155 160
Glu Ser Met Thr Ser Ala Thr Phe Arg Asp Thr Asn Glu Asp Glu Ile
165 170 175
Tyr Ala Phe Phe Gly Ile Leu Val Met Thr Ala Val Arg Lys Asp Asn
180 185 190
His Met Ser Thr Asp Asp Leu Phe Asp Arg Ser Leu Ser Met Val Tyr
195 200 205
Val Ser Val Met Ser Arg Asp Arg Phe Asp Phe Leu Ile Arg Cys Leu
210 215 220
Arg Met Asp Asp Lys Ser Ile Arg Pro Thr Leu Arg Glu Asn Asp Val
225 230 235 240
Phe Thr Pro Val Arg Lys Ile Trp Asp Leu Phe Ile His Gln Cys Ile
245 250 255
Gln Asn Tyr Thr Pro Gly Ala His Leu Thr Ile Asp Glu Gln Leu Leu
260 265 270
Gly Phe Arg Gly Arg Cys Pro Phe Arg Val Tyr Ile Pro Asn Lys Pro
275 280 285
Ser Lys Tyr Gly Ile Lys Ile Leu Met Met Cys Asp Ser Gly Thr Lys
290 295 300
Tyr Met Ile Asn Gly Met Pro Tyr Leu Gly Arg Gly Thr Gln Thr Asn
305 310 315 320
Gly Val Pro Leu Gly Glu Tyr Tyr Val Lys Glu Leu Ser Lys Pro Val
325 330 335
His Gly Ser Cys Arg Asn Ile Thr Cys Asp Asn Trp Phe Thr Ser Ile
340 345 350
Pro Leu Ala Lys Asn Leu Leu Gln Glu Pro Tyr Lys Leu Thr Ile Val
355 360 365
Gly Thr Val Arg Ser Asn Lys Arg Glu Ile Pro Glu Val Leu Lys Asn
370 375 380
Ser Arg Ser Arg Pro Val Gly Thr Ser Met Phe Cys Phe Asp Gly Pro
385 390 395 400
Leu Thr Leu Val Ser Tyr Lys Pro Lys Pro Ala Lys Met Val Tyr Leu
405 410 415
Leu Ser Ser Cys Asp Glu Asp Ala Ser Ile Asn Glu Ser Thr Gly Lys
420 425 430
Pro Gln Met Val Met Tyr Tyr Asn Gln Thr Lys Gly Gly Val Asp Thr
435 440 445
Leu Asp Gln Met Cys Ser Val Met Thr Cys Ser Arg Lys Thr Asn Arg
450 455 460
Trp Pro Met Ala Leu Leu Tyr Gly Met Ile Asn Ile Ala Cys Ile Asn
465 470 475 480
Ser Phe Ile Ile Tyr Ser His Asn Val Ser Ser Lys Gly Glu Lys Val
485 490 495
Gln Ser Arg Lys Lys Phe Met Arg Asn Leu Tyr Met Gly Leu Thr Ser
500 505 510
Ser Phe Met Arg Lys Arg Leu Glu Ala Pro Thr Leu Lys Arg Tyr Leu
515 520 525
Arg Asp Asn Ile Ser Asn Ile Leu Pro Lys Glu Val Pro Gly Thr Ser
530 535 540
Asp Asp Ser Thr Glu Glu Pro Val Met Lys Lys Arg Thr Tyr Cys Thr
545 550 555 560
Tyr Cys Pro Ser Lys Ile Arg Arg Lys Ala Ser Ala Ser Cys Lys Xaa
565 570 575
Cys Lys Lys Val Ile Cys Arg Glu His Asn Xaa Asp Met Cys Gln Ser
580 585 590
Cys Phe
<210> 17
<211> 594
<212> PRT
<213> Artificial Sequence
<220>
<223> Modified hyperactive PiggyBac with mutations involved in Zn2+
binding
<220>
<221> misc_feature
<222> (586)..(586)
<223> Wherein amino acid can be His, any naturally occuring amino acid
<400> 17
Met Gly Ser Ser Leu Asp Asp Glu His Ile Leu Ser Ala Leu Leu Gln
1 5 10 15
Ser Asp Asp Glu Leu Val Gly Glu Asp Ser Asp Ser Glu Val Ser Asp
20 25 30
His Val Ser Glu Asp Asp Val Gln Ser Asp Thr Glu Glu Ala Phe Ile
35 40 45
Asp Glu Val His Glu Val Gln Pro Thr Ser Ser Gly Ser Glu Ile Leu
50 55 60
Asp Glu Gln Asn Val Ile Glu Gln Pro Gly Ser Ser Leu Ala Ser Asn
65 70 75 80
Arg Ile Leu Thr Leu Pro Gln Arg Thr Ile Arg Gly Lys Asn Lys His
85 90 95
Cys Trp Ser Thr Ser Lys Pro Thr Arg Arg Ser Arg Val Ser Ala Leu
100 105 110
Asn Ile Val Arg Ser Gln Arg Gly Pro Thr Arg Met Cys Arg Asn Ile
115 120 125
Tyr Asp Pro Leu Leu Cys Phe Lys Leu Phe Phe Thr Asp Glu Ile Ile
130 135 140
Ser Glu Ile Val Lys Trp Thr Asn Ala Glu Ile Ser Leu Lys Arg Arg
145 150 155 160
Glu Ser Met Thr Ser Ala Thr Phe Arg Asp Thr Asn Glu Asp Glu Ile
165 170 175
Tyr Ala Phe Phe Gly Ile Leu Val Met Thr Ala Val Arg Lys Asp Asn
180 185 190
His Met Ser Thr Asp Asp Leu Phe Asp Arg Ser Leu Ser Met Val Tyr
195 200 205
Val Ser Val Met Ser Arg Asp Arg Phe Asp Phe Leu Ile Arg Cys Leu
210 215 220
Arg Met Asp Asp Lys Ser Ile Arg Pro Thr Leu Arg Glu Asn Asp Val
225 230 235 240
Phe Thr Pro Val Arg Lys Ile Trp Asp Leu Phe Ile His Gln Cys Ile
245 250 255
Gln Asn Tyr Thr Pro Gly Ala His Leu Thr Ile Asp Glu Gln Leu Leu
260 265 270
Gly Phe Arg Gly Arg Cys Pro Phe Arg Val Tyr Ile Pro Asn Lys Pro
275 280 285
Ser Lys Tyr Gly Ile Lys Ile Leu Met Met Cys Asp Ser Gly Thr Lys
290 295 300
Tyr Met Ile Asn Gly Met Pro Tyr Leu Gly Arg Gly Thr Gln Thr Asn
305 310 315 320
Gly Val Pro Leu Gly Glu Tyr Tyr Val Lys Glu Leu Ser Lys Pro Val
325 330 335
His Gly Ser Cys Arg Asn Ile Thr Cys Asp Asn Trp Phe Thr Ser Ile
340 345 350
Pro Leu Ala Lys Asn Leu Leu Gln Glu Pro Tyr Lys Leu Thr Ile Val
355 360 365
Gly Thr Val Arg Ser Asn Lys Arg Glu Ile Pro Glu Val Leu Lys Asn
370 375 380
Ser Arg Ser Arg Pro Val Gly Thr Ser Met Phe Cys Phe Asp Gly Pro
385 390 395 400
Leu Thr Leu Val Ser Tyr Lys Pro Lys Pro Ala Lys Met Val Tyr Leu
405 410 415
Leu Ser Ser Cys Asp Glu Asp Ala Ser Ile Asn Glu Ser Thr Gly Lys
420 425 430
Pro Gln Met Val Met Tyr Tyr Asn Gln Thr Lys Gly Gly Val Asp Thr
435 440 445
Leu Asp Gln Met Cys Ser Val Met Thr Cys Ser Arg Lys Thr Asn Arg
450 455 460
Trp Pro Met Ala Leu Leu Tyr Gly Met Ile Asn Ile Ala Cys Ile Asn
465 470 475 480
Ser Phe Ile Ile Tyr Ser His Asn Val Ser Ser Lys Gly Glu Lys Val
485 490 495
Gln Ser Arg Lys Lys Phe Met Arg Asn Leu Tyr Met Gly Leu Thr Ser
500 505 510
Ser Phe Met Arg Lys Arg Leu Glu Ala Pro Thr Leu Lys Arg Tyr Leu
515 520 525
Arg Asp Asn Ile Ser Asn Ile Leu Pro Lys Glu Val Pro Gly Thr Ser
530 535 540
Asp Asp Ser Thr Glu Glu Pro Val Met Lys Lys Arg Thr Tyr Cys Thr
545 550 555 560
Tyr Cys Pro Ser Lys Ile Arg Arg Lys Ala Ser Ala Ser Cys Lys Lys
565 570 575
Cys Lys Lys Val Ile Cys Arg Glu His Asn Ile Asp Met Cys Gln Ser
580 585 590
Cys Phe
<210> 18
<211> 594
<212> PRT
<213> Artificial Sequence
<220>
<223> Modified hyperactive PiggyBac with mutations that are involved in
integration
<220>
<221> MISC_FEATURE
<222> (315)..(315)
<223> Wherein amino acid can be Arg, Ala
<220>
<221> MISC_FEATURE
<222> (341)..(341)
<223> Wherein amino acid can be Arg, Ala
<220>
<221> MISC_FEATURE
<222> (372)..(372)
<223> Wherein amino acid can be Arg, Ala
<220>
<221> MISC_FEATURE
<222> (375)..(372)
<223> Wherein amino acid can be Lys, Ala
<400> 18
Met Gly Ser Ser Leu Asp Asp Glu His Ile Leu Ser Ala Leu Leu Gln
1 5 10 15
Ser Asp Asp Glu Leu Val Gly Glu Asp Ser Asp Ser Glu Val Ser Asp
20 25 30
His Val Ser Glu Asp Asp Val Gln Ser Asp Thr Glu Glu Ala Phe Ile
35 40 45
Asp Glu Val His Glu Val Gln Pro Thr Ser Ser Gly Ser Glu Ile Leu
50 55 60
Asp Glu Gln Asn Val Ile Glu Gln Pro Gly Ser Ser Leu Ala Ser Asn
65 70 75 80
Arg Ile Leu Thr Leu Pro Gln Arg Thr Ile Arg Gly Lys Asn Lys His
85 90 95
Cys Trp Ser Thr Ser Lys Pro Thr Arg Arg Ser Arg Val Ser Ala Leu
100 105 110
Asn Ile Val Arg Ser Gln Arg Gly Pro Thr Arg Met Cys Arg Asn Ile
115 120 125
Tyr Asp Pro Leu Leu Cys Phe Lys Leu Phe Phe Thr Asp Glu Ile Ile
130 135 140
Ser Glu Ile Val Lys Trp Thr Asn Ala Glu Ile Ser Leu Lys Arg Arg
145 150 155 160
Glu Ser Met Thr Ser Ala Thr Phe Arg Asp Thr Asn Glu Asp Glu Ile
165 170 175
Tyr Ala Phe Phe Gly Ile Leu Val Met Thr Ala Val Arg Lys Asp Asn
180 185 190
His Met Ser Thr Asp Asp Leu Phe Asp Arg Ser Leu Ser Met Val Tyr
195 200 205
Val Ser Val Met Ser Arg Asp Arg Phe Asp Phe Leu Ile Arg Cys Leu
210 215 220
Arg Met Asp Asp Lys Ser Ile Arg Pro Thr Leu Arg Glu Asn Asp Val
225 230 235 240
Phe Thr Pro Val Arg Lys Ile Trp Asp Leu Phe Ile His Gln Cys Ile
245 250 255
Gln Asn Tyr Thr Pro Gly Ala His Leu Thr Ile Asp Glu Gln Leu Leu
260 265 270
Gly Phe Arg Gly Arg Cys Pro Phe Arg Val Tyr Ile Pro Asn Lys Pro
275 280 285
Ser Lys Tyr Gly Ile Lys Ile Leu Met Met Cys Asp Ser Gly Thr Lys
290 295 300
Tyr Met Ile Asn Gly Met Pro Tyr Leu Gly Arg Gly Thr Gln Thr Asn
305 310 315 320
Gly Val Pro Leu Gly Glu Tyr Tyr Val Lys Glu Leu Ser Lys Pro Val
325 330 335
His Gly Ser Cys Arg Asn Ile Thr Cys Asp Asn Trp Phe Thr Ser Ile
340 345 350
Pro Leu Ala Lys Asn Leu Leu Gln Glu Pro Tyr Lys Leu Thr Ile Val
355 360 365
Gly Thr Val Arg Ser Asn Lys Arg Glu Ile Pro Glu Val Leu Lys Asn
370 375 380
Ser Arg Ser Arg Pro Val Gly Thr Ser Met Phe Cys Phe Asp Gly Pro
385 390 395 400
Leu Thr Leu Val Ser Tyr Lys Pro Lys Pro Ala Lys Met Val Tyr Leu
405 410 415
Leu Ser Ser Cys Asp Glu Asp Ala Ser Ile Asn Glu Ser Thr Gly Lys
420 425 430
Pro Gln Met Val Met Tyr Tyr Asn Gln Thr Lys Gly Gly Val Asp Thr
435 440 445
Leu Asp Gln Met Cys Ser Val Met Thr Cys Ser Arg Lys Thr Asn Arg
450 455 460
Trp Pro Met Ala Leu Leu Tyr Gly Met Ile Asn Ile Ala Cys Ile Asn
465 470 475 480
Ser Phe Ile Ile Tyr Ser His Asn Val Ser Ser Lys Gly Glu Lys Val
485 490 495
Gln Ser Arg Lys Lys Phe Met Arg Asn Leu Tyr Met Gly Leu Thr Ser
500 505 510
Ser Phe Met Arg Lys Arg Leu Glu Ala Pro Thr Leu Lys Arg Tyr Leu
515 520 525
Arg Asp Asn Ile Ser Asn Ile Leu Pro Lys Glu Val Pro Gly Thr Ser
530 535 540
Asp Asp Ser Thr Glu Glu Pro Val Met Lys Lys Arg Thr Tyr Cys Thr
545 550 555 560
Tyr Cys Pro Ser Lys Ile Arg Arg Lys Ala Ser Ala Ser Cys Lys Lys
565 570 575
Cys Lys Lys Val Ile Cys Arg Glu His Asn Ile Asp Met Cys Gln Ser
580 585 590
Cys Phe
<210> 19
<211> 897
<212> PRT
<213> Artificial Sequence
<220>
<223> Cas9 from Corynebacterium ulcerans
<400> 19
Met Thr Asn Ala Val Ala Asn His His Val Leu Trp Ala Lys Phe Asp
1 5 10 15
Asn Val Ser Glu Pro Tyr Pro Leu Leu Ala His Leu Leu Asp Thr Ala
20 25 30
Thr Ala Ala Thr Cys Leu Phe Asn His Trp Leu Arg Lys Gly Leu Arg
35 40 45
Asp Arg Leu Ser Thr Glu Leu Gly Pro Asp Ala Glu Lys Ile Leu Gly
50 55 60
Phe Val Ala Gly Ile His Asp Leu Gly Lys Ala Asn Pro Tyr Phe Gln
65 70 75 80
Ala Gln Arg Arg Asn Lys Lys Glu Glu Trp Ile Thr Leu Arg Asp Ala
85 90 95
Ile Gln Lys Ala Gly Phe Pro Leu Ser Asn Gly Thr Ser Ala Leu Phe
100 105 110
Glu Glu Thr Lys Glu Lys Arg Arg His Glu Asn Ile Thr Leu Ser Ile
115 120 125
Leu Gly Trp Glu Ile Thr Lys Phe Leu Gln Val Lys Asp Val Trp Pro
130 135 140
Gln Leu Ala Ile Ile Gly His His Gly Asn Phe Ser Ala Pro Gly Phe
145 150 155 160
Leu Ser Asp Glu Asp Asp Leu Glu Asp Ile Glu Asp Ile Phe Asp Asp
165 170 175
Asn Gly Trp Ser Pro Thr His Glu Leu Leu Val Ser Ser Leu Leu Gln
180 185 190
Ala Val Gly Leu Glu Lys Gln Pro Glu Ile Lys His Ile Ser Pro Ala
195 200 205
Ser Ala Ile Leu Ile Ser Gly Leu Val Val Leu Ala Asp Arg Ile Ala
210 215 220
Ser Gln Ser Glu Met Ala Ser Asp Gly Leu Gln Ala Leu Gln Lys Glu
225 230 235 240
Glu Leu Phe Phe His Gln Pro Glu Lys Trp Ile Ala Asn Arg Lys Ala
245 250 255
Phe Cys Arg Glu Ile Ile Glu Asn Thr Val Gly Thr Tyr His Pro Trp
260 265 270
Glu Ser Glu Ala Ala Gly Ile Arg Ala Val Leu Gly Asp Tyr Glu Pro
275 280 285
Arg Phe Thr Gln Lys Ala Ala Leu Asn Ala Gly Asp Gly Leu Phe Asn
290 295 300
Val Met Glu Thr Thr Gly Ala Gly Lys Thr Glu Ala Ala Leu Leu Arg
305 310 315 320
His Val Lys Arg Lys Glu Arg Leu Leu Phe Phe Leu Pro Thr Gln Ala
325 330 335
Thr Thr Asn Ala Ile Met Asp Arg Ile Gly Lys Ile Phe Asp Gly Thr
340 345 350
Pro Asn Val Ala Ser Leu Ala His Gly Leu Ala Val Thr Glu Asp Phe
355 360 365
Tyr Ala His Pro Ile Leu Pro Val Gln Gly Ser Ser Asp Asp Ala Asn
370 375 380
Tyr Lys Asp Asn Gly Gly Leu Tyr Pro Thr Glu Phe Val Arg Ser Ala
385 390 395 400
Gly Thr Pro Arg Leu Leu Ala Pro Val Cys Val Gly Thr Ile Asp Gln
405 410 415
Ala Leu Met Gly Ala Leu Pro Ser Lys Phe Asn His Leu Arg Leu Leu
420 425 430
Ala Leu Ala Asn Ala His Val Val Val Asp Glu Val His Thr Met Asp
435 440 445
Gln Tyr Gln Ser Glu Leu Met Ser Gly Leu Leu Glu Trp Trp Ser Ala
450 455 460
Thr Asp Thr Pro Val Thr Leu Leu Thr Ala Thr Met Pro Ala Trp Gln
465 470 475 480
Arg Glu Lys Phe His Leu Ser Tyr Thr Gly Lys Asp Pro His Phe Lys
485 490 495
Gly Val Phe Pro Ser Leu Glu Asp Trp Ser Thr Pro Ser Lys Asn Thr
500 505 510
Glu Thr Ser Gln Glu Asn Ile Pro Thr Glu Ala Phe Thr Ile Pro Ile
515 520 525
Asn Ile Asp Lys Ile Ala His Asn Glu Ile Val Asp Ser His Val Gln
530 535 540
Trp Val Ile Glu Gln Arg Lys Leu Phe Pro Gln Ala Arg Ile Gly Ile
545 550 555 560
Ile Cys Asn Thr Val Gly Arg Ala Gln Ser Ile Ala Glu Ala Leu Ala
565 570 575
His Glu Ser Pro Ile Val Leu His Ser Arg Met Thr Ala Gly His Arg
580 585 590
Lys Glu Ala Ala Thr Lys Leu Glu Gln Ala Ile Gly Lys Lys Gly Thr
595 600 605
Ala Asn Ala Thr Leu Val Ile Gly Thr Gln Ala Ile Glu Ala Ser Leu
610 615 620
Asp Ile Asp Leu Asp Leu Leu Arg Thr Glu Leu Cys Pro Ala Pro Ser
625 630 635 640
Leu Ile Gln Arg Ala Gly Arg Leu Trp Arg Arg Leu Asp Pro Gln Arg
645 650 655
Glu Val Arg Val Pro Gly Met Val Gly Lys Lys Leu Thr Ile Ala Val
660 665 670
Val Asp Ser Pro Ser Thr Gly Gln Thr Leu Pro Tyr Leu Arg Ser Gln
675 680 685
Leu Tyr Arg Val Glu Ser Trp Leu Lys Gln Arg Asp Arg Ile Glu Phe
690 695 700
Pro Ala Asp Ile Gln Asp Phe Ile Asp Ala Thr Thr Pro Gly Leu Gln
705 710 715 720
Glu Leu Phe Gln Lys Val Ser Leu Pro Glu Asp Cys Gly Ser Ala Glu
725 730 735
Glu Arg Glu Ala Leu Ala Asp Asp Tyr Leu Asn Glu Val Ala Ser Trp
740 745 750
Val Thr Lys Gln Arg Gln Ala Gly Thr Ser Arg Ile Asp Phe Ala Lys
755 760 765
His Gly Lys Pro Arg Gln Val Leu Ala Ser Asp Cys Val Val Glu Asp
770 775 780
Phe Leu Gln Ile Thr Ser Ala Asn Asn Leu Glu Glu Ser Ala Thr Arg
785 790 795 800
Leu Ile Asp Tyr Pro Ser Ile Ser Ala Ile Leu Cys Asp Pro Thr Gly
805 810 815
Thr Ile Pro Gly Ala Trp Thr Asp Ser Val Glu Lys Leu Ile Ala Ile
820 825 830
Ser Ala Lys Asp Ser Glu Ser Leu Arg Arg Ala Leu Arg Ala Ser Ile
835 840 845
Ser Ile Pro His Ser Lys Lys Phe Leu Pro Ile Thr Ser Arg Glu Ile
850 855 860
Pro Leu Ser Glu Ala Lys Thr Leu Leu Ser Gly Tyr Ser Ala Val His
865 870 875 880
Ile Gln Pro Asp Glu Tyr Asp Leu Gln Ser Gly Leu Lys Gly Pro Gln
885 890 895
Lys
<210> 20
<211> 876
<212> PRT
<213> Artificial Sequence
<220>
<223> Cas9 from Corynebacterium diphtheria
<400> 20
Met Asn Pro His Glu Glu Leu Trp Ala Lys Gln Lys Gly Leu Ala Lys
1 5 10 15
Pro Tyr Pro Leu Leu Ala His Leu Leu Asp Ser Ala Ala Val Ala Gly
20 25 30
Ala Leu Trp Asp His Trp Leu Arg Gln Asp Leu Arg Gln Met Phe Ile
35 40 45
Glu Glu Leu Gly Ser Asn Ala Arg Glu Ile Ile Gln Phe Val Val Gly
50 55 60
Ser His Asp Ile Gly Lys Ala Thr Pro Leu Phe Gln Tyr Gln Lys Ala
65 70 75 80
Gln Lys Gly Glu Val Trp Asp Ser Ile Arg Tyr Ala Ile Asp Arg Thr
85 90 95
Gly Arg Tyr Gln Lys Pro Leu Pro Ser Ser Tyr Leu Val Lys Lys Thr
100 105 110
Ser Gly Gly Pro Asn Arg His Glu Gln Trp Ser Ser Phe Ala Ser Lys
115 120 125
Asn Glu Tyr Leu Lys Pro Ser Ala Ala Ala Lys Glu Asn Trp Ile Gly
130 135 140
Leu Ala Ile Gly Gly His His Gly Arg Phe Glu Pro Val Gly Tyr Gly
145 150 155 160
Arg His Gln Arg Lys Ala Ala Glu Asp Leu Ala Lys Ser Gly Trp Ser
165 170 175
Ala Ala Gln Gln Asp Leu Leu Arg Ala Leu Glu Lys Ala Ser Gly Ile
180 185 190
Thr Arg Ala Ser Leu Pro Ser Glu Leu Ser Pro Glu Leu Thr Leu Val
195 200 205
Leu Ser Gly Leu Thr Ile Leu Ala Asp Arg Ile Ser Ser Thr Glu Ser
210 215 220
Phe Val Ile Thr Gly Ala Arg Met Ile Asp Asp Gly Thr Leu His Leu
225 230 235 240
Ala Thr Pro Ile Asp Trp Leu Lys Thr Arg Lys Leu Asp Ser Glu Lys
245 250 255
His Val Ala Lys Thr Val Gly Ile Tyr His Gly Trp Asn Asn His Glu
260 265 270
Ser Ala Ile His Ser Ile Leu Lys Gly Tyr Asp Pro Arg Pro Leu Gln
275 280 285
Thr Ile Ala Leu Gln Asn Gln Val Gly Leu Leu Asn Leu Met Ala Pro
290 295 300
Thr Gly Asn Gly Lys Thr Glu Ala Ala Ile Leu Arg His Ser Leu Lys
305 310 315 320
Glu Asn Asp Arg Leu Ile Phe Leu Leu Pro Thr Gln Ala Thr Ser Asn
325 330 335
Ala Ile Met Arg Arg Val Gln Gly Ile Tyr Ser Asp Thr Pro Asn Ala
340 345 350
Ala Ala Leu Ala His Ser Leu Ala Ser Val Glu Asp Phe Tyr Gln Thr
355 360 365
Pro Leu Ser Val Phe Asp Asp His Tyr Asp Pro Ser Lys Glu Gln Phe
370 375 380
Glu Ser Ser Met Ser Gly Gly Leu Tyr Pro Ser Ser Phe Val Cys Ser
385 390 395 400
Gly Ala Ala Arg Leu Leu Ala Pro Ile Cys Ile Gly Thr Val Asp Gln
405 410 415
Ala Leu Ala Thr Ala Leu Pro Gly Lys Trp Ile His Leu Arg Ile Leu
420 425 430
Ala Leu Ala Asn Ala His Ile Val Ile Asp Glu Val His Thr Leu Asp
435 440 445
His Tyr Gln Thr Ala Leu Leu Glu Asn Ile Leu Pro Ile Leu Ala Lys
450 455 460
Leu Lys Thr Lys Ile Thr Phe Leu Thr Ala Thr Met Pro Ser Trp Gln
465 470 475 480
Arg Thr Lys Leu Leu Thr Ala Tyr Gly Gly Glu Asp Leu Gln Ile Pro
485 490 495
Pro Thr Val Phe Pro Ala Ala Glu Thr Val Leu Pro Gly Gln Phe Asn
500 505 510
Arg Thr Leu Ile Asp Ser Asp Ser Thr Thr Ile Asp Phe Thr Met Glu
515 520 525
Glu Thr Ser Tyr Asp His Leu Val Glu Ser His Val Lys Trp His Gln
530 535 540
Thr Thr Arg Leu Asn Ala Pro His Ala Arg Ile Gly Leu Ile Cys Asn
545 550 555 560
Thr Val Lys Arg Ala Gln Glu Ile Ala Ala Ala Leu Glu Lys Thr Asn
565 570 575
Asp Arg Ile Val Leu Leu His Ser Arg Met Thr Thr Glu His Arg Arg
580 585 590
Arg Ser Ala Glu Leu Leu Glu Ser Leu Leu Gly Pro Asn Gly Asn Arg
595 600 605
Lys Thr Ile Thr Val Val Gly Thr Gln Ala Ile Glu Ala Ser Leu Asp
610 615 620
Ile Asp Leu Asp Ile Leu Arg Thr Glu Leu Cys Pro Ala Pro Ser Leu
625 630 635 640
Val Gln Arg Ala Gly Arg Val Trp Arg Arg Asn Asp Pro Tyr Arg Ser
645 650 655
Ser Arg Ile Thr Ala Asp His Lys Pro Ile Ser Val Val Phe Ile Ala
660 665 670
Glu Ala Lys Asp Trp Gln Val Leu Pro Tyr Leu Arg Ala Glu Thr Ser
675 680 685
Arg Thr Gln Arg Trp Leu Glu Lys His Asn Gln Met Phe Leu Pro Gln
690 695 700
Met Ala Gln Glu Phe Ile Asp Ala Ala Thr Val Asp Leu Asp Thr Ala
705 710 715 720
Thr Ser Glu Met Asp Leu Asp Ala Leu Ala Leu Met Gly Ile His Leu
725 730 735
Met Lys Ala Asp Gly Ala Lys Ala Arg Ile Gln Asp Val Leu Asn Ser
740 745 750
Asp Ser Lys Val Ser Asp Phe Ala Leu Leu Thr Ser Lys Asn Glu Ile
755 760 765
Asp Glu Ala Gln Thr Arg Leu Ile Glu Glu Gly Thr His Leu Arg Ile
770 775 780
Ile Leu Gly Asp Glu Asn Glu Ser Ile Pro Gly Gly Trp Lys His Gly
785 790 795 800
Leu Ser Ser Leu Leu Lys Leu Lys Ala Ser Asp Arg Glu Ser Leu Arg
805 810 815
Thr Ala Leu Leu Ala Ser Ile Pro Leu Leu Val Ser Glu Lys Gln Lys
820 825 830
Gln Leu Leu Tyr Gln His Asn Leu Val Pro Leu Ser Ser Ser Lys Thr
835 840 845
Val Leu Ala Gly Phe Tyr Phe Leu Pro Lys Ala Gln Asn Phe Tyr Ser
850 855 860
Lys Asn Leu Gly Phe Ile Trp Pro Glu Glu Lys Asp
865 870 875
<210> 21
<211> 773
<212> PRT
<213> Artificial Sequence
<220>
<223> Cas9 from Spiroplasma syrphidicola
<400> 21
Met Asn Tyr Lys Lys Leu Ile Leu Gly Leu Asp Leu Gly Ile Ala Ser
1 5 10 15
Cys Gly Trp Ala Val Thr Gly Gln Met Glu Asp Gly Asn Trp Val Leu
20 25 30
Asp Asp Phe Gly Val Arg Leu Phe Gln Thr Pro Glu Asn Ser Lys Asp
35 40 45
Gly Thr Thr Asn Ala Ala Ala Arg Arg Leu Lys Arg Gly Ala Arg Arg
50 55 60
Leu Ile Lys Arg Arg Lys Asn Arg Ile Lys Asp Leu Lys Asn Leu Phe
65 70 75 80
Glu Lys Ile Asn Phe Ile Asn Lys Ala Ser Leu Asp Lys Tyr Ile Asn
85 90 95
Glu His Ser Ala Thr Asn Leu Val Glu Asp Phe Asn Arg His Glu Leu
100 105 110
Tyr Asn Pro Tyr Phe Leu Arg Ser Ile Gly Ile Thr Glu Lys Leu Thr
115 120 125
Arg Glu Glu Leu Val Trp Ser Leu Ile His Ile Ala Asn Arg Arg Gly
130 135 140
Tyr Lys Asn Lys Phe Ala Phe Asp Ile Glu Gly Asp Gly Lys Lys Arg
145 150 155 160
Glu Thr Lys Leu Asp Glu Ala Ile Ser Asn Ala Leu Ile Ser Ser Asn
165 170 175
Leu Thr Ile Ser Gln Glu Ile Val Arg Asn Lys Lys Phe Arg Asp Ala
180 185 190
Lys Asn Lys Lys Ala Leu Leu Val Arg Asn Lys Gly Gly Lys Glu Gly
195 200 205
Glu Asn Asn Phe Gln Phe Leu Phe Ala Arg Asp Asp Tyr Lys Lys Glu
210 215 220
Val Asp Leu Leu Leu Ala Lys Gln Ala Lys Phe Tyr Pro Glu Leu Thr
225 230 235 240
Glu Glu Ile Arg Ala Lys Ala Ala Asp Ile Ile Phe Arg Gln Arg Asp
245 250 255
Phe Glu Asp Gly Pro Gly Pro Lys Lys Gln Glu Leu Arg Glu Ile Tyr
260 265 270
Lys Lys Glu Asn Lys Gln Phe Ser Lys Asn Phe Thr Gln Leu Glu Gly
275 280 285
Arg Cys Thr Phe Leu Arg Glu Leu Ser Val Gly Tyr Lys Ser Ser Ile
290 295 300
Leu Phe Asp Leu Phe His Ile Ile Ser Glu Val Ser Lys Ile Ser Lys
305 310 315 320
Tyr Ile Glu Glu Asn Asp Gln Leu Ala Gln Asp Ile Ile Ser Ser Phe
325 330 335
Leu Tyr Asn Glu Ala Gly Lys Lys Gly Lys Thr Leu Leu Lys Glu Ile
340 345 350
Leu Lys Lys His His Ile Asn Asp Asp Ile Phe Asp Thr Asn Ala Tyr
355 360 365
Lys Asn Ile Asp Phe Lys Thr Asn Tyr Leu Asn Leu Leu Lys Glu Val
370 375 380
Phe Gly Asn Asp Val Leu Lys Asn Leu Ser Leu Asn Arg Leu Glu Asp
385 390 395 400
Asn Ile Tyr His Gln Leu Gly Phe Ile Ile His Thr Asn Ile Thr Pro
405 410 415
Glu Arg Lys Glu Lys Ala Ile Asn Gln Trp Leu Leu Glu Asn Asn Ile
420 425 430
Ile Leu Ala Lys Glu Lys Leu Asn Ile Leu Leu Lys Pro Asn Ser Ser
435 440 445
Ile Ser Thr Thr Val Lys Thr Ser Phe Lys Trp Met Ser Ile Ala Ile
450 455 460
Ser Asn Phe Leu Lys Gly Ile Pro Tyr Gly Lys Phe Gln Ala Gln Phe
465 470 475 480
Ile Lys Glu Asp Asn Phe Lys Leu Pro Glu Ser Tyr Ala Lys Gln Tyr
485 490 495
Gln Lys Tyr Leu Thr Gly Glu Lys Thr Phe Glu Met Phe Ala Pro Ile
500 505 510
Ile Asp Pro Asp Leu Trp Arg Asn Pro Ile Val Phe Arg Ala Ile Asn
515 520 525
Gln Ala Arg Lys Val Ile Lys Lys Leu Phe Glu Lys Tyr Thr Phe Ile
530 535 540
Asp Gln Ile Asn Ile Glu Leu Thr Arg Glu Met Gly Leu Ser Phe Ser
545 550 555 560
Asp Arg Lys Lys Val Lys Glu Arg Gln Asp Asp Ser Leu Lys Glu Asn
565 570 575
Ala Lys Ala Lys Glu Phe Leu Met Ala Asn Gly Ile Ile Val Asn Asp
580 585 590
Thr Asn Val Leu Lys Tyr Lys Leu Trp Ile Gln Gln Asn Lys Lys Ser
595 600 605
Leu Tyr Ser Gly Lys Glu Ile Thr Ile Ala Asp Leu Gly Ala Ser Asn
610 615 620
Val Leu Gln Ile Asp His Ile Ile Pro Tyr Ser Lys Leu Ala Asp Asp
625 630 635 640
Ser Phe Asn Asn Lys Val Leu Val Phe Ser Lys Glu Asn Gln Glu Lys
645 650 655
Gly Asn Gln Phe Ala Asp Gln Tyr Val Lys Ser Leu Gly Thr Glu Asn
660 665 670
Tyr Asn Asn Tyr Lys Lys Arg Val Asn Tyr Leu Leu Phe Gln Asn Gln
675 680 685
Ile Asn Gln Lys Lys Ala Glu Tyr Leu Leu Cys Ser Asn Gln Asn Glu
690 695 700
Glu Ile Leu Asn Asp Phe Val Ser Arg Asn Leu Asn Asp Thr Arg Tyr
705 710 715 720
Ile Thr Arg Tyr Val Thr Asn Trp Leu Lys Ala Glu Phe Glu Leu Gln
725 730 735
Ser Arg Phe Gly Leu Ala Lys Pro Lys Ile Met Thr Leu Asn Gly Ala
740 745 750
Ile Thr Ser Arg Phe Arg Arg Thr Trp Leu Arg Asn Ser Pro Trp Gly
755 760 765
Leu Glu Lys Lys Ser
770
<210> 22
<211> 1380
<212> PRT
<213> Artificial Sequence
<220>
<223> Cas9 from Prevotella intermedia
<400> 22
Met Lys Arg Ile Leu Gly Leu Asp Leu Gly Thr Thr Ser Ile Gly Trp
1 5 10 15
Ala Leu Val Asn Glu Ala Glu Asn Asn Asn Glu Ala Ser Ser Ile Val
20 25 30
Arg Leu Gly Val Arg Val Asn Pro Leu Thr Val Asp Glu Lys Ser Asn
35 40 45
Phe Glu Lys Gly Lys Ala Ile Thr Thr Asn Ala Asp Arg Gln Leu Arg
50 55 60
His Gly Ala Arg Ile Asn Leu Gln Arg Tyr Lys Leu Arg Arg Gln Asn
65 70 75 80
Leu His Asp Cys Leu Gln Lys Gln Gly Trp Leu Gly Thr Glu Ala Met
85 90 95
Tyr Glu Glu Gly Lys Ala Ser Thr Phe Glu Thr Tyr Lys Leu Arg Ala
100 105 110
Lys Ala Ala Glu Glu Glu Ile Ser Leu His Glu Phe Ala Arg Val Leu
115 120 125
Phe Met Leu Asn Lys Lys Arg Gly Tyr Lys Ser Asn Arg Lys Ala Asn
130 135 140
Asn Lys Glu Asp Gly Gln Leu Phe Asp Gly Met Thr Ile Ala Lys Lys
145 150 155 160
Leu Tyr Glu Glu His Leu Thr Pro Ala Glu Tyr Ser Leu Gln Leu Leu
165 170 175
Asn Lys Gly Lys Lys Phe Thr Gln Gly Tyr Tyr Arg Ser Asp Leu Asn
180 185 190
Ala Glu Leu Glu Arg Ile Trp Asp Glu Gln Lys Lys Tyr Tyr Pro Glu
195 200 205
Ile Leu Thr Asp Glu Phe Lys Gln Gln Leu Glu Gly Lys Thr Lys Thr
210 215 220
Asn Thr Ser Lys Ile Phe Leu Ala Lys Tyr Gly Ile Tyr Ser Ala Asp
225 230 235 240
Leu Lys Gly Leu Asp Arg Lys Phe Gln Pro Leu Lys Trp Arg Val Glu
245 250 255
Ala Leu Gln Gln Gln Val Asp Lys Glu Val Leu Ala Phe Val Ile Ser
260 265 270
Asp Leu Lys Gly Gln Ile Ala Asn Thr Ser Gly Leu Leu Gly Ala Ile
275 280 285
Ser Asp Arg Ser Lys Glu Leu Tyr Phe Asn Lys Gln Thr Val Gly Gln
290 295 300
Tyr Leu Trp Ala Ser Leu Glu Glu Asn Pro His Ile Ser Ile Lys Asn
305 310 315 320
Lys Pro Phe Tyr Arg Gln Asp Tyr Leu Asp Glu Phe Glu Lys Ile Trp
325 330 335
Glu Thr Gln Ala Ala Phe His Lys Gln Leu Thr Pro Glu Leu Lys Gln
340 345 350
Glu Ile Arg Asp Ile Ile Ile Phe Tyr Gln Arg Pro Leu Lys Ser Lys
355 360 365
Lys Ser Leu Ile Ser Val Cys Glu Leu Glu Gln Arg Lys Val Lys Ala
370 375 380
Thr Ile Asp Gly Lys Glu Lys Glu Ile Thr Ile Gly Pro Lys Val Ala
385 390 395 400
Pro Lys Ser Ser Pro Val Phe Gln Glu Phe Arg Ile Trp Gln Asn Leu
405 410 415
Asn Asn Val Leu Leu Ile Asp Asn Asp Thr Asn Glu Lys Arg Pro Leu
420 425 430
Asp Glu Val Glu Arg Asn Leu Leu Tyr Lys Glu Leu Ser Ile Lys Ala
435 440 445
Lys Leu Ser Lys Thr Glu Ala Leu Lys Ile Leu Asn Lys Lys Gly Lys
450 455 460
Gln Trp Asp Leu Asn Tyr Arg Glu Leu Glu Gly Asn Arg Thr Gln Ala
465 470 475 480
Ile Leu Phe Asp Cys Tyr Asn Arg Ile Ile Thr Leu Thr Gly His Glu
485 490 495
Glu Cys Asp Phe Lys Lys Ile Lys Ala Ser Glu Ile Arg His Tyr Val
500 505 510
Ser Thr Ile Phe Lys Asn Leu Gly Phe Ser Thr Glu Ile Leu Asp Phe
515 520 525
Asp Pro Ser Leu Lys Lys His Glu Leu Glu Lys Gln Pro Met Tyr Gln
530 535 540
Leu Trp His Leu Leu Tyr Ser Tyr Glu Ser Asp Asn Ser Arg Thr Gly
545 550 555 560
Asn Glu Ser Leu Leu Arg Lys Leu Glu Thr Thr Phe Gly Phe Pro Glu
565 570 575
Glu Tyr Ala Thr Val Leu Cys Asp Val Val Phe Glu Glu Asp Tyr Gly
580 585 590
Asn Leu Ser Val Lys Ala Met Arg Glu Ile Leu Pro Tyr Leu Gln Ala
595 600 605
Gly Asn Asp Tyr Ser Gln Ala Cys Ala Tyr Ala Gly Tyr Asn His Ser
610 615 620
Arg His Ser Leu Thr Lys Glu Glu Leu Asp Gln Lys Val Tyr Lys Glu
625 630 635 640
Arg Leu Glu Leu Leu Pro Lys Asn Ser Leu Arg Asn Pro Val Val Glu
645 650 655
Lys Ile Leu Asn Gln Met Ile Asn Val Ile Asn Ala Ile Ile Asp Glu
660 665 670
Tyr Gly Lys Pro Asp Glu Ile Arg Ile Glu Met Ala Arg Glu Leu Lys
675 680 685
Ser Ser Ala Ala Asp Arg Lys Lys Thr Thr His Ala Ile Ser Gln Gly
690 695 700
Asn Ala Glu Asn Gln Arg Ile Arg Glu Ile Leu Glu Lys Glu Phe Ser
705 710 715 720
Leu Ser Tyr Ile Ser Arg Asn Asp Ile Ile Lys Tyr Lys Leu Tyr Glu
725 730 735
Glu Leu Glu Pro Asn Tyr Tyr Lys Thr Leu Tyr Ser Asp Thr Tyr Ile
740 745 750
Thr Lys Asp Lys Leu Phe Ser Lys Asp Phe Asp Ile Glu His Ile Ile
755 760 765
Pro Lys Ala Arg Leu Phe Asp Asp Ser Phe Ser Asn Lys Thr Leu Glu
770 775 780
Ala Arg Asn Ile Asn Leu Glu Lys Ser Asn Lys Thr Ala Phe Asp Phe
785 790 795 800
Ile Lys Glu Lys Tyr Gly Glu Asp Gly Ala Glu Ala Tyr Lys Lys Lys
805 810 815
Leu Asp Met Leu Leu Glu Asn Asp Ala Ile Ser Arg Pro Lys Tyr Asn
820 825 830
Asn Leu Leu Arg Ala Glu Ala Asp Ile Pro Ser Asp Phe Ile Asn Arg
835 840 845
Asp Leu Arg Asn Thr Gln Tyr Ile Ala Lys Lys Ala Cys Glu Ile Leu
850 855 860
Gly Glu Leu Val Lys Thr Val Thr Pro Thr Thr Gly Lys Ile Thr Asn
865 870 875 880
Arg Leu Arg Glu Asp Trp Gln Leu Val Asp Val Met Lys Glu Leu Asn
885 890 895
Phe Glu Lys Tyr Glu Lys Leu Gly Leu Thr Glu Ile Val Glu Asp Arg
900 905 910
Asp Gly Arg Lys Ile Lys Arg Ile Lys Asp Trp Thr Lys Arg Asn Asp
915 920 925
His Arg His His Ala Met Asp Ala Leu Ala Ile Ala Phe Thr Lys Pro
930 935 940
Ser Phe Ile Gln Tyr Leu Asn Asn Leu Asn Ala Arg Ser Asn Lys Gly
945 950 955 960
Asp Ser Ile Tyr Ala Ile Glu Asn Lys Glu Leu His Tyr Glu Glu Gly
965 970 975
Lys Leu Arg Phe Asn Ala Pro Ile Pro Val Asn Glu Phe Arg Ala Glu
980 985 990
Ala Lys Arg His Leu Ser Ala Ile Leu Val Ser Ile Lys Ala Lys Asn
995 1000 1005
Lys Val Met Thr Gln Asn Val Asn Lys Ile Lys Thr Lys His Gly
1010 1015 1020
Ile Ile Lys Lys Ile Gln Leu Thr Pro Arg Gly Pro Leu His Asn
1025 1030 1035
Glu Thr Ile Tyr Gly Thr Lys Met Arg Pro Ile Ile Lys Met Val
1040 1045 1050
Lys Val Gly Ala Ala Leu Asp Glu Ala Thr Ile Asn Lys Val Ser
1055 1060 1065
Ser Pro Ala Ile Arg Glu Ala Leu Leu Lys Arg Leu Asn Glu Tyr
1070 1075 1080
Ser Gly Asn Ala Lys Lys Ala Phe Thr Gly Lys Asn Thr Leu Glu
1085 1090 1095
Lys Asn Pro Ile Tyr Leu Asn Ala Gly Arg Thr Lys Thr Val Pro
1100 1105 1110
Ser Leu Val Lys Thr Val Glu Trp Glu Ser Phe His Pro Thr Arg
1115 1120 1125
Lys Leu Ile Asp Lys Asp Leu Asn Val Asp Lys Val Val Asp Lys
1130 1135 1140
Gly Ile Arg Glu Ile Leu Lys Ala Arg Leu Glu Glu Phe Asn Gly
1145 1150 1155
Asp Ala Lys Lys Ala Phe Ser Asn Leu Glu Glu Asn Pro Ile Tyr
1160 1165 1170
Leu Asp Glu Ala Lys Lys Ile Ala Leu Lys Arg Val Ser Ile Glu
1175 1180 1185
Gly Val Leu Ser Ala Ile Pro Leu His Thr Leu Lys Asn Gln Ala
1190 1195 1200
Gly Lys Pro Ile Thr Gly Lys Asp Gly Lys Pro Val Leu Gly Asn
1205 1210 1215
Tyr Val Gln Thr Ser Asn Asn His His Ile Ala Phe Tyr Tyr Asp
1220 1225 1230
Glu Asp Gly Asn Leu Gln Asp Asn Ala Val Ser Phe Phe Glu Ala
1235 1240 1245
Ala Glu Arg Lys Ser Gln Gly Ile Pro Val Ile Asp Lys Asp Tyr
1250 1255 1260
Asn Arg Asp Lys Gly Trp Arg Phe Leu Phe Thr Met Lys Gln Asn
1265 1270 1275
Glu Tyr Phe Val Phe Pro Asn Glu Ala Thr Gly Phe Ile Pro Ser
1280 1285 1290
Glu Val Asp Leu Thr Asp Glu Ala Asn Tyr Gly Ile Ile Ser Pro
1295 1300 1305
Asn Leu Tyr Arg Val Gln Lys Val Ser Arg Ile Asp Lys Gly Thr
1310 1315 1320
Ser Ala Ser Arg Asp Tyr Trp Phe Arg His His Leu Glu Thr Ile
1325 1330 1335
Leu Asn Asp Asp Ala Lys Leu Lys Asn Leu Ala Phe Lys Arg Ile
1340 1345 1350
Arg Gly Leu Leu Glu Leu Lys Asp Ile Ile Lys Val Arg Ile Asn
1355 1360 1365
Ser Thr Gly Lys Ile Val Ala Val Gly Glu Tyr Asp
1370 1375 1380
<210> 23
<211> 535
<212> PRT
<213> Artificial Sequence
<220>
<223> Cas9 from Spiroplasma taiwanense
<400> 23
Met Trp Ser Arg Lys Ile Leu Lys Ala Gly Ser Arg Leu Phe Asp Glu
1 5 10 15
Ala Asn Leu Ser Asp Lys Ile Ala Ser Lys Arg Arg Glu Gln Arg Gly
20 25 30
Arg Arg Arg Asn Leu Arg Arg Lys Ile Thr Trp Lys Gln Asp Leu Ile
35 40 45
Asn Leu Phe Val Lys Tyr Asn Phe Leu Gln Lys Glu Asn Asp Phe Tyr
50 55 60
Glu Leu Asp Phe Asn Phe Asp Leu Leu Glu Leu Arg Lys Lys Ala Ile
65 70 75 80
Asn Ser Lys Ile Glu Leu Glu Gln Leu Leu Ile Ile Leu Phe Asn Tyr
85 90 95
Ile Lys His Arg Gly Ser Phe Asn Tyr Arg Glu Asp Leu Ser Glu Leu
100 105 110
Lys Asn Ile Ser Gln Glu Glu Leu Glu Thr Ser Ser Glu Phe Lys Leu
115 120 125
Pro Val Asp Ile Gln Phe Glu Leu Lys Glu Glu Asn Asn Lys Phe Arg
130 135 140
Glu Ile Asn Asn Glu Lys Ser Leu Ile Asn His Glu Trp Tyr Val Lys
145 150 155 160
Glu Ile Asn Leu Ile Leu Asp Ala Gln Ile Glu Asn Lys Leu Ile Asn
165 170 175
Leu Asp Phe Lys Lys Asp Tyr Leu Lys Leu Phe Asn Arg Lys Arg Glu
180 185 190
Tyr Tyr Asp Gly Pro Gly Pro Lys Asp Lys Asn Leu Leu Asn Pro Ser
195 200 205
Lys Tyr Gly Trp Lys Asn Gln Glu Glu Phe Phe Asp Arg Phe Ala Gly
210 215 220
Lys Asp Thr Tyr Asp Ser Lys Glu Gln Arg Ala Pro Lys His Ser Leu
225 230 235 240
Thr Ser Tyr Leu Phe Asn Ile Leu Asn Asp Leu Asn Asn Leu Ser Ile
245 250 255
Asn Gly Asp Arg Asn Gln Leu Thr Tyr Glu Asn Lys Lys Asp Leu Ile
260 265 270
Asn Leu Thr Leu Ile Asn Gln Lys Glu Lys Ala Glu Asn Ile Thr Leu
275 280 285
Lys Lys Ile Ala Lys Tyr Leu Lys Ile Asn Glu Lys Asn Ile Thr Gly
290 295 300
Tyr Arg Leu Lys Pro Asn Ser Asn Glu Ser Ile Phe Thr Val Phe Glu
305 310 315 320
Ser Ala Asn Lys Met Arg Ser Ile Leu Val Lys Asn Asn Lys Ser Ile
325 330 335
Asp Phe Ile Cys Leu Glu Asn Ile Asp Lys Ile Asp Lys Ile Val Asp
340 345 350
Ile Leu Thr Lys Tyr Gln Ser Ile Glu Asp Lys Ser Leu Lys Leu Glu
355 360 365
Glu Leu Asn Phe Asp Phe Phe Asp Lys Glu Thr Cys Glu Lys Leu Ala
370 375 380
Val Ile Ser Leu Thr Gly Thr His Ala Leu Ser Lys Lys Thr Met Ser
385 390 395 400
Lys Leu Ile Glu Glu Met Phe His Asp Asn Leu Asn His Met Glu Ala
405 410 415
Leu Ala Lys Leu Lys Ile Lys Pro Asp Tyr Lys Leu Lys Val Asp Leu
420 425 430
Thr Asn Phe Lys Thr Ile Pro Ile Leu Arg Glu Lys Ile Asn Glu Met
435 440 445
Tyr Ile Ser Pro Val Val Lys Arg Ala Leu Ile Glu Ser Leu Lys Ile
450 455 460
Ile Lys Glu Leu Glu Arg His Phe Lys Asp Phe Glu Ile Lys Asp Ile
465 470 475 480
Val Ile Glu Met Ala Lys Lys Asn Ser Ala Glu Lys Lys Gln Phe Ile
485 490 495
Ser Lys Ile Gln Arg Gln Asn Val Asp Leu Val Lys Lys Leu Ser Asn
500 505 510
Asp Tyr Ser Leu Asp Glu Asn Lys Leu Asn Phe Lys Met Lys Glu Lys
515 520 525
Phe Leu Leu Leu Ser Glu Gln
530 535
<210> 24
<211> 1281
<212> PRT
<213> Artificial Sequence
<220>
<223> Cas9 from Streptococcus iniae
<400> 24
Met Arg Lys Pro Tyr Ser Ile Gly Leu Asp Ile Gly Thr Asn Ser Val
1 5 10 15
Gly Trp Ala Val Ile Thr Asp Asp Tyr Lys Val Pro Ser Lys Lys Met
20 25 30
Arg Ile Gln Gly Thr Thr Asp Arg Thr Ser Ile Lys Lys Asn Leu Ile
35 40 45
Gly Ala Leu Leu Phe Asp Asn Gly Glu Thr Ala Glu Ala Thr Arg Leu
50 55 60
Lys Arg Thr Thr Arg Arg Arg Tyr Thr Arg Arg Lys Tyr Arg Ile Lys
65 70 75 80
Glu Leu Gln Lys Ile Phe Ser Ser Glu Met Asn Glu Leu Asp Ile Ala
85 90 95
Phe Phe Pro Arg Leu Ser Glu Ser Phe Leu Val Ser Asp Asp Lys Glu
100 105 110
Phe Glu Asn His Pro Ile Phe Gly Asn Leu Lys Asp Glu Ile Thr Tyr
115 120 125
His Asn Asp Tyr Pro Thr Ile Tyr His Leu Arg Gln Thr Leu Ala Asp
130 135 140
Ser Asp Gln Lys Ala Asp Leu Arg Leu Ile Tyr Leu Ala Leu Ala His
145 150 155 160
Ile Ile Lys Phe Arg Gly His Phe Leu Ile Glu Gly Asn Leu Asp Ser
165 170 175
Glu Asn Thr Asp Val His Val Leu Phe Leu Asn Leu Val Asn Ile Tyr
180 185 190
Asn Asn Leu Phe Glu Glu Asp Ile Val Glu Thr Ala Ser Ile Asp Ala
195 200 205
Glu Lys Ile Leu Thr Ser Lys Thr Ser Lys Ser Arg Arg Leu Glu Asn
210 215 220
Leu Ile Ala Glu Ile Pro Asn Gln Lys Arg Asn Met Leu Phe Gly Asn
225 230 235 240
Leu Val Ser Leu Ala Leu Gly Leu Thr Pro Asn Phe Lys Thr Asn Phe
245 250 255
Glu Leu Leu Glu Asp Ala Lys Leu Gln Ile Ser Lys Asp Ser Tyr Glu
260 265 270
Glu Asp Leu Asp Asn Leu Leu Ala Gln Ile Gly Asp Gln Tyr Ala Asp
275 280 285
Leu Phe Ile Ala Ala Lys Lys Leu Ser Asp Ala Ile Leu Leu Ser Asp
290 295 300
Ile Ile Thr Val Lys Gly Ala Ser Thr Lys Ala Pro Leu Ser Ala Ser
305 310 315 320
Met Val Gln Arg Tyr Glu Glu His Gln Gln Asp Leu Ala Leu Leu Lys
325 330 335
Asn Leu Val Lys Lys Gln Ile Pro Glu Lys Tyr Lys Glu Ile Phe Asp
340 345 350
Asn Lys Glu Lys Asn Gly Tyr Ala Gly Tyr Ile Asp Gly Lys Thr Ser
355 360 365
Gln Glu Glu Phe Tyr Lys Tyr Ile Lys Pro Ile Leu Leu Lys Leu Asp
370 375 380
Gly Thr Glu Lys Leu Ile Ser Lys Leu Glu Arg Glu Asp Phe Leu Arg
385 390 395 400
Lys Gln Arg Thr Phe Asp Asn Gly Ser Ile Pro His Gln Ile His Leu
405 410 415
Asn Glu Leu Lys Ala Ile Ile Arg Arg Gln Glu Lys Phe Tyr Pro Phe
420 425 430
Leu Lys Glu Asn Gln Lys Lys Ile Glu Lys Leu Phe Thr Phe Lys Ile
435 440 445
Pro Tyr Tyr Val Gly Pro Leu Ala Asn Gly Gln Ser Ser Phe Ala Trp
450 455 460
Leu Lys Arg Gln Ser Asn Glu Ser Ile Thr Pro Trp Asn Phe Glu Glu
465 470 475 480
Val Val Asp Gln Glu Ala Ser Ala Arg Ala Phe Ile Glu Arg Met Thr
485 490 495
Asn Phe Asp Thr Tyr Leu Pro Glu Glu Lys Val Leu Pro Lys His Ser
500 505 510
Pro Leu Tyr Glu Met Phe Met Val Tyr Asn Glu Leu Thr Lys Val Lys
515 520 525
Tyr Gln Thr Glu Gly Met Lys Arg Pro Val Phe Leu Ser Ser Glu Asp
530 535 540
Lys Glu Glu Ile Val Asn Leu Leu Phe Lys Lys Glu Arg Lys Val Thr
545 550 555 560
Val Lys Gln Leu Lys Glu Glu Tyr Phe Ser Lys Met Lys Cys Phe His
565 570 575
Thr Val Thr Ile Leu Gly Val Glu Asp Arg Phe Asn Ala Ser Leu Gly
580 585 590
Thr Tyr His Asp Leu Leu Lys Ile Phe Lys Asp Lys Ala Phe Leu Asp
595 600 605
Asp Glu Ala Asn Gln Asp Ile Leu Glu Glu Ile Val Trp Thr Leu Thr
610 615 620
Leu Phe Glu Asp Gln Ala Met Ile Glu Arg Arg Leu Val Lys Tyr Ala
625 630 635 640
Asp Val Phe Glu Lys Ser Val Leu Lys Lys Leu Lys Lys Arg His Tyr
645 650 655
Thr Gly Trp Gly Arg Leu Ser Gln Lys Leu Ile Asn Gly Ile Lys Asp
660 665 670
Lys Gln Thr Gly Lys Thr Ile Leu Gly Phe Leu Lys Asp Asp Gly Val
675 680 685
Ala Asn Arg Asn Phe Met Gln Leu Ile Asn Asp Ser Ser Leu Asp Phe
690 695 700
Ala Lys Ile Ile Lys Asn Glu Gln Glu Lys Thr Ile Lys Asn Glu Ser
705 710 715 720
Leu Glu Glu Thr Ile Ala Asn Leu Ala Gly Ser Pro Ala Ile Lys Lys
725 730 735
Gly Ile Leu Gln Ser Ile Lys Ile Val Asp Glu Ile Val Lys Ile Met
740 745 750
Gly Gln Asn Pro Asp Asn Ile Val Ile Glu Met Ala Arg Glu Asn Gln
755 760 765
Ser Thr Met Gln Gly Ile Lys Asn Ser Arg Gln Arg Leu Arg Lys Leu
770 775 780
Glu Glu Val His Lys Asn Thr Gly Ser Lys Ile Leu Lys Glu Tyr Asn
785 790 795 800
Val Ser Asn Thr Gln Leu Gln Ser Asp Arg Leu Tyr Leu Tyr Leu Leu
805 810 815
Gln Asp Gly Lys Asp Met Tyr Thr Gly Lys Glu Leu Asp Tyr Asp Asn
820 825 830
Leu Ser Gln Tyr Asp Ile Asp His Ile Ile Pro Gln Ser Phe Ile Lys
835 840 845
Asp Asn Ser Ile Asp Asn Thr Val Leu Thr Thr Gln Ala Ser Asn Arg
850 855 860
Gly Lys Ser Asp Asn Val Pro Asn Ile Glu Thr Val Asn Lys Met Lys
865 870 875 880
Ser Phe Trp Tyr Lys Gln Leu Lys Ser Gly Ala Ile Ser Gln Arg Lys
885 890 895
Phe Asp His Leu Thr Lys Ala Glu Arg Gly Ala Leu Ser Asp Phe Asp
900 905 910
Lys Ala Gly Phe Ile Lys Arg Gln Leu Val Glu Thr Arg Gln Ile Thr
915 920 925
Lys His Val Ala Gln Ile Leu Asp Ser Arg Phe Asn Ser Asn Leu Thr
930 935 940
Glu Asp Ser Lys Ser Asn Arg Asn Val Lys Ile Ile Thr Leu Lys Ser
945 950 955 960
Lys Met Val Ser Asp Phe Arg Lys Asp Phe Gly Phe Tyr Lys Leu Arg
965 970 975
Glu Val Asn Asp Tyr His His Ala Gln Asp Ala Tyr Leu Asn Ala Val
980 985 990
Val Gly Thr Ala Leu Leu Lys Lys Tyr Pro Lys Leu Glu Ala Glu Phe
995 1000 1005
Val Tyr Gly Asp Tyr Lys His Tyr Asp Leu Ala Lys Leu Met Ile
1010 1015 1020
Gln Pro Asp Ser Ser Leu Gly Lys Ala Thr Thr Arg Met Phe Phe
1025 1030 1035
Tyr Ser Asn Leu Met Asn Phe Phe Lys Lys Glu Ile Lys Leu Ala
1040 1045 1050
Asp Asp Thr Ile Phe Thr Arg Pro Gln Ile Glu Val Asn Thr Glu
1055 1060 1065
Thr Gly Glu Ile Val Trp Asp Lys Val Lys Asp Met Gln Thr Ile
1070 1075 1080
Arg Lys Val Met Ser Tyr Pro Gln Val Asn Ile Val Met Lys Thr
1085 1090 1095
Glu Val Gln Thr Gly Gly Phe Ser Lys Glu Ser Ile Trp Pro Lys
1100 1105 1110
Gly Asp Ser Asp Lys Leu Ile Ala Arg Lys Lys Ser Trp Asp Pro
1115 1120 1125
Lys Lys Tyr Gly Gly Phe Asp Ser Pro Ile Ile Ala Tyr Ser Val
1130 1135 1140
Leu Val Val Ala Lys Ile Ala Lys Gly Lys Thr Gln Lys Leu Lys
1145 1150 1155
Thr Ile Lys Glu Leu Val Gly Ile Lys Ile Met Glu Gln Asp Glu
1160 1165 1170
Phe Glu Lys Asp Pro Ile Ala Phe Leu Glu Lys Lys Gly Tyr Gln
1175 1180 1185
Asp Ile Gln Thr Ser Ser Ile Ile Lys Leu Pro Lys Tyr Ser Leu
1190 1195 1200
Phe Glu Leu Glu Asn Gly Arg Lys Arg Leu Leu Ala Ser Ala Lys
1205 1210 1215
Glu Leu Gln Lys Gly Asn Glu Leu Ala Leu Pro Asn Lys Tyr Val
1220 1225 1230
Lys Phe Leu Tyr Leu Ala Ser His Tyr Thr Lys Phe Thr Gly Lys
1235 1240 1245
Glu Glu Asp Arg Glu Lys Lys Arg Ser Tyr Val Glu Ser His Leu
1250 1255 1260
Tyr Tyr Phe Asp Val Arg Leu Ser Gln Val Phe Arg Val Thr Asn
1265 1270 1275
Val Glu Phe
1280
<210> 25
<211> 1352
<212> PRT
<213> Artificial Sequence
<220>
<223> Cas9 from Belliella baltica
<400> 25
Met Lys Lys Ile Leu Gly Leu Asp Leu Gly Thr Thr Ser Ile Gly Trp
1 5 10 15
Ala Phe Ile Lys Glu Pro Glu Lys Asp Val Val Gly Ser Glu Ile Val
20 25 30
Asp Met Gly Val Arg Ile Val Pro Leu Ser Ser Asp Glu Glu Asn Asp
35 40 45
Phe Ala Lys Gly Asn Thr Ile Ser Ile Asn Ala Asp Arg Thr Leu Lys
50 55 60
Arg Gly Ala Arg Arg Asn Leu Gln Arg Phe Lys Gln Arg Arg Asn Ala
65 70 75 80
Leu Leu Glu Ile Phe Lys Glu Lys Lys Leu Ile Ser Thr Asn Phe Lys
85 90 95
Tyr Ala Glu Asp Gly Pro Ser Ser Thr Phe Ser Thr Leu Asn Leu Arg
100 105 110
Ala Lys Ala Ala Lys Glu Lys Ile Glu Leu Gln Asp Leu Val Lys Val
115 120 125
Leu Leu Gln Ile Asn Lys Lys Arg Gly Tyr Lys Ser Ser Arg Lys Ala
130 135 140
Lys Ser Glu Glu Asp Asp Gly Ser Ala Ile Asp Ser Met Gly Ile Ala
145 150 155 160
Lys Glu Leu Tyr Glu Asn Asp Leu Thr Pro Gly Gln Trp Val Tyr Glu
165 170 175
Ala Leu Gln Lys Gly Arg Lys Asn Val Pro Asp Phe Tyr Arg Ser Asp
180 185 190
Leu Gln Glu Glu Phe Lys Lys Ile Val Asn Tyr Gln Ser Glu Phe Phe
195 200 205
Pro Asp Ile Phe Asn Ala Ser Phe Val Glu Asp Trp Met Gly Lys Ala
210 215 220
Ser Thr Pro Thr Lys Gln Tyr Phe Asn Lys Lys Gly Val Gln Leu Ala
225 230 235 240
Glu Asn Lys Gly Lys Arg Glu Glu Arg Arg Leu Gln Glu Tyr Lys Trp
245 250 255
Arg Ala Glu Ala Val Asn Phe Lys Ile Asp Leu Ser Glu Ile Ala Leu
260 265 270
Ile Leu Ser Gln Ile Asn Ser Gln Ile Ser Asn Ser Ser Gly Tyr Leu
275 280 285
Gly Ala Ile Ser Asp Arg Ser Lys Glu Leu Tyr Phe Lys Asn Leu Thr
290 295 300
Val Gly Gln Tyr Leu Tyr Gln Gln Ile Lys Lys Asn Pro His Thr Arg
305 310 315 320
Leu Lys Gly Gln Val Phe Tyr Arg Gln Asp Tyr Leu Asp Glu Phe Glu
325 330 335
Arg Ile Trp Ser Val Gln Ser Ser Phe Tyr Pro Gln Leu Asn Asp Ala
340 345 350
Leu Lys Arg Glu Val Arg Asp Ile Thr Ile Phe Phe Gln Arg Arg Leu
355 360 365
Lys Ser Gln Lys His Leu Ile Ser Asn Cys Glu Phe Glu Asp His His
370 375 380
Lys Val Val Pro Lys Ser His Pro Val Phe Gln Glu Phe Arg Ile Trp
385 390 395 400
Gln Asn Leu Asn Asn Leu Leu Leu Ile Lys Lys Asp Asn Leu Asn Glu
405 410 415
Lys Phe Asp Leu Glu Leu Glu Ser Lys Ile Ala Leu Ala Asn Glu Leu
420 425 430
Ala Phe Lys Arg Glu Leu Asn Val Lys Asp Ala Leu Lys Ile Leu Gly
435 440 445
Leu Lys Pro Asn Glu Trp Glu Phe Asn Phe Thr Lys Ile Glu Gly Asn
450 455 460
Arg Thr Asn Gln Ala Phe Phe Asp Ala Phe Ala Lys Ile Ile Glu Leu
465 470 475 480
Glu Asp Gly Glu Pro Ile Asp Leu Gly Asp Leu Lys Ala Asp Asp Ile
485 490 495
Leu Asp Gln Phe Ser Glu Ala Phe Leu Arg Ile Gly Ile Asp Thr Glu
500 505 510
Leu Leu Gln Val Asn Ser Asp Ile Glu Gly Ala Glu Tyr Glu Lys Gln
515 520 525
Ser Tyr Ile Gln Phe Trp His Leu Leu Tyr Ser Ser Glu Asp Asp Gln
530 535 540
Lys Leu Lys Leu Asn Leu Ile Arg Lys Phe Gly Phe Lys Pro Glu His
545 550 555 560
Ala Lys Ile Leu Ala Ser Ile Ser Leu Gln Asp Asp His Ala Ser Leu
565 570 575
Ser Ser Arg Ala Ile Lys Lys Ile Leu Pro His Leu Gln Ser Gly Leu
580 585 590
Ile Tyr Asp Lys Ala Cys Thr Tyr Ala Gly Tyr Asn His Ser Ser Ser
595 600 605
Phe Thr Lys Asp Glu Asn Glu Lys Arg Glu Leu Arg Ala Glu Leu Glu
610 615 620
Leu Leu Lys Lys Asn Ser Leu Arg Asn Pro Val Val Glu Lys Ile Leu
625 630 635 640
Asn Gln Met Ile Asn Val Val Asn Ala Ile Leu Lys Asp Pro Glu Leu
645 650 655
Gly Arg Pro Asp Glu Ile Arg Val Glu Met Ala Arg Glu Leu Lys Ala
660 665 670
Asn Ala Glu Gln Arg Lys Asn Met Thr Ser Asn Ile Ala Ser Ala Thr
675 680 685
Arg Asp His Asp Lys Tyr Arg Glu Ile Leu Lys Ser Glu Phe Gly Leu
690 695 700
Lys Arg Val Thr Lys Asn Asp Leu Leu Arg Tyr Lys Leu Trp Leu Glu
705 710 715 720
Thr Asp Gly Ile Ser Leu Tyr Thr Gly Lys Pro Ile Glu Ala Ser Lys
725 730 735
Leu Phe Ser Lys Glu Tyr Asp Ile Glu His Ile Ile Pro Lys Ala Arg
740 745 750
Leu Phe Asp Asp Ser Phe Ser Asn Lys Thr Ile Cys Glu Arg Gln Leu
755 760 765
Asn Ile Asp Lys Ala Asn Val Thr Ala Phe Ser Phe Leu Gln Asn Lys
770 775 780
Leu Ser Ala Asp Glu Phe Glu Gln Tyr Gln Ser Arg Val Lys Ser Leu
785 790 795 800
Tyr Gly Lys Leu Ser Lys Ala Lys Ile Gln Lys Leu Leu Met Ala Asn
805 810 815
Asp Lys Ile Pro Glu Asp Phe Ile Ala Arg Gln Leu Gln Glu Thr Arg
820 825 830
Tyr Ile Ser Lys Lys Ala Lys Glu Ile Leu Phe Glu Ile Ser Arg Arg
835 840 845
Val Ser Val Thr Thr Gly Thr Ile Thr Asp Lys Leu Arg Glu Asp Trp
850 855 860
Gly Leu Val Glu Ile Met Lys Glu Leu Asn Trp Glu Lys Tyr Asp Lys
865 870 875 880
Leu Gly Leu Thr Tyr Thr Ile Glu Gly Lys His Gly Glu Arg Leu Asn
885 890 895
Lys Ile Lys Asp Trp Ser Lys Arg Asn Asp His Arg His His Ala Met
900 905 910
Asp Ala Leu Thr Val Ala Leu Thr Lys Pro Ala Tyr Ile Gln Tyr Leu
915 920 925
Asn Asn Leu Asn Ala Lys Gly Leu Asn Asn Lys Lys Gly Thr Glu Val
930 935 940
Phe Ala Ile Glu Gln Lys Tyr Leu Lys Arg Glu Asn Gly Lys Leu Cys
945 950 955 960
Phe Ile Pro Pro Ile Glu Asn Ile Arg Ser Glu Ala Lys Lys His Leu
965 970 975
Ser Arg Ile Leu Val Ser Tyr Lys Ala Lys Asn Lys Val Val Thr Ile
980 985 990
Asn Lys Asn Lys Thr Lys Ser Lys Ala Gly Leu Asn Glu Gln Ile Ala
995 1000 1005
Leu Thr Pro Arg Gly Gln Leu His Lys Glu Thr Val Tyr Gly Lys
1010 1015 1020
Ser Phe His Tyr Ser Thr Lys Phe Glu Lys Ile Gly Ala Ser Phe
1025 1030 1035
Asn Val Gln Lys Ile Asn Thr Val Ala Lys Lys Glu Glu Arg Glu
1040 1045 1050
Ala Leu Leu Lys Arg Leu Ala Glu Asn Gly Asn Asp Pro Lys Lys
1055 1060 1065
Ala Phe Thr Gly Lys Asn Thr Leu Asn Lys Met Pro Ile Tyr Leu
1070 1075 1080
Asp Leu Gly Lys Asn Ile Lys Leu Ser Glu Lys Val Lys Thr Val
1085 1090 1095
Val Leu Glu Gln Asn Tyr Thr Ile Arg Lys Asn Ile Asp Pro Asp
1100 1105 1110
Leu Lys Val Asp Lys Val Ile Asp Val Gly Ile Lys Arg Ile Leu
1115 1120 1125
Glu Ser Arg Leu Glu Glu Phe Gly Gly Asn Ala Lys Leu Ala Phe
1130 1135 1140
Ser Asn Leu Glu Glu Asn Pro Ile Trp Leu Asn Lys Glu Lys Gly
1145 1150 1155
Ile Ser Ile Lys Arg Val Lys Ile Ser Gly Val Ser Asn Val Glu
1160 1165 1170
Ser Leu His Val Lys Lys Asp His Phe Gly Glu Pro Ile Leu Asp
1175 1180 1185
Gln Glu Gly Asn Glu Ile Pro Val Asp Phe Val Ser Thr Gly Asn
1190 1195 1200
Asn His His Val Ala Ile Tyr Glu Asp Glu Asn Gly Asn Leu Gln
1205 1210 1215
Glu Glu Val Val Ser Phe Phe Glu Ala Val Val Arg Gln Asn Gln
1220 1225 1230
Gly Leu Pro Ile Ile Lys Lys Asn His Thr Leu Gly Trp Lys Phe
1235 1240 1245
Leu Phe Thr Leu Lys Gln Asn Glu Tyr Phe Val Phe Pro Ser Asp
1250 1255 1260
Asp Phe Val Pro Ala Asp Val Asp Leu Met Asp Glu Gln Asn Tyr
1265 1270 1275
His Leu Ile Ser Pro Asn Leu Phe Arg Val Gln Lys Ile Ala Arg
1280 1285 1290
Lys Asn Tyr Val Phe Asn Asn His Leu Glu Thr Lys Ala Val Asp
1295 1300 1305
Asn Asp Leu Leu Lys Ser Lys Lys Glu Leu Ser Lys Ile Thr Tyr
1310 1315 1320
His Phe Tyr Gln Thr Pro Glu His Leu Arg Gly Ile Ile Lys Ile
1325 1330 1335
Arg Ile Asn His Leu Gly Lys Ile Ile Gln Ile Gly Glu Tyr
1340 1345 1350
<210> 26
<211> 1509
<212> PRT
<213> Artificial Sequence
<220>
<223> Cas9 from Psychroflexus torquisI
<400> 26
Met Lys Arg Ile Leu Gly Leu Asp Leu Gly Thr Asn Ser Ile Gly Trp
1 5 10 15
Ser Leu Ile Glu His Asp Phe Lys Asn Lys Gln Gly Gln Ile Glu Gly
20 25 30
Leu Gly Val Arg Ile Ile Pro Met Ser Gln Glu Ile Leu Gly Lys Phe
35 40 45
Asp Ala Gly Gln Ser Ile Ser Gln Thr Ala Asp Arg Thr Lys Tyr Arg
50 55 60
Gly Val Arg Arg Leu Tyr Gln Arg Asp Asn Leu Arg Arg Glu Arg Leu
65 70 75 80
His Arg Val Leu Lys Ile Leu Asp Phe Leu Pro Lys His Tyr Ser Glu
85 90 95
Ser Ile Asp Phe Gln Asp Lys Val Gly Gln Phe Lys Pro Lys Gln Glu
100 105 110
Val Lys Leu Asn Tyr Arg Lys Asn Glu Lys Asn Lys His Glu Phe Val
115 120 125
Phe Met Asn Ser Phe Ile Glu Met Val Ser Glu Phe Lys Asn Ala Gln
130 135 140
Pro Glu Leu Phe Tyr Asn Lys Gly Asn Gly Glu Glu Thr Lys Ile Pro
145 150 155 160
Tyr Asp Trp Thr Leu Tyr Tyr Leu Arg Lys Lys Ala Leu Thr Gln Gln
165 170 175
Ile Thr Lys Glu Glu Leu Ala Trp Leu Ile Leu Asn Phe Asn Gln Lys
180 185 190
Arg Gly Tyr Tyr Gln Leu Arg Gly Glu Asp Ile Asp Glu Asp Lys Asn
195 200 205
Lys Lys Tyr Met Gln Leu Lys Val Asn Asn Leu Ile Asp Ser Gly Ala
210 215 220
Lys Val Lys Gly Lys Val Leu Tyr Asn Val Ile Phe Asp Asn Gly Trp
225 230 235 240
Lys Tyr Glu Lys Gln Ile Val Asn Lys Asp Glu Trp Glu Gly Arg Thr
245 250 255
Lys Glu Phe Ile Ile Thr Thr Lys Thr Leu Lys Asn Gly Asn Ile Lys
260 265 270
Arg Thr Tyr Lys Ala Val Asp Ser Glu Ile Asp Trp Ala Ala Ile Lys
275 280 285
Ala Lys Thr Glu Gln Asp Ile Asn Lys Ala Asn Lys Thr Val Gly Glu
290 295 300
Tyr Ile Tyr Glu Ser Leu Leu Asp Asn Pro Ser Gln Lys Ile Arg Gly
305 310 315 320
Lys Leu Val Lys Thr Ile Glu Arg Lys Phe Tyr Lys Glu Glu Phe Glu
325 330 335
Lys Leu Leu Ser Lys Gln Ile Glu Leu Gln Pro Glu Leu Phe Asn Glu
340 345 350
Ser Leu Tyr Lys Ala Cys Ile Lys Glu Leu Tyr Pro Arg Asn Glu Asn
355 360 365
His Gln Ser Asn Asn Lys Lys Gln Gly Phe Glu Tyr Leu Phe Thr Glu
370 375 380
Asp Ile Ile Phe Tyr Gln Arg Pro Leu Lys Ser Gln Lys Ser Asn Ile
385 390 395 400
Ser Gly Cys Gln Phe Glu His Lys Ile Tyr Lys Gln Lys Asn Lys Lys
405 410 415
Thr Gly Lys Leu Glu Leu Ile Lys Glu Pro Ile Lys Thr Ile Ser Arg
420 425 430
Ser His Pro Leu Phe Gln Glu Phe Arg Ile Trp Gln Trp Leu Gln Asn
435 440 445
Leu Lys Ile Tyr Asn Lys Glu Lys Ile Glu Asn Gly Lys Leu Glu Asp
450 455 460
Val Thr Thr Gln Leu Leu Pro Asn Asn Glu Ala Tyr Val Thr Leu Phe
465 470 475 480
Asp Phe Leu Asn Thr Lys Lys Glu Leu Glu Gln Lys Gln Phe Ile Glu
485 490 495
Tyr Phe Val Lys Lys Lys Leu Ile Asp Lys Lys Glu Lys Glu His Phe
500 505 510
Arg Trp Asn Phe Val Glu Asp Lys Lys Tyr Pro Phe Ser Glu Thr Arg
515 520 525
Ala Gln Phe Leu Ser Arg Leu Ala Lys Val Lys Gly Ile Lys Asn Thr
530 535 540
Glu Asp Phe Leu Asn Lys Asn Thr Gln Val Gly Ser Lys Glu Asn Ser
545 550 555 560
Pro Phe Ile Lys Arg Ile Glu Gln Leu Trp His Ile Ile Tyr Ser Val
565 570 575
Ser Asp Leu Lys Glu Tyr Glu Lys Ala Leu Glu Lys Phe Ala Glu Lys
580 585 590
His Asn Leu Glu Lys Asp Ser Phe Leu Lys Asn Phe Lys Lys Phe Pro
595 600 605
Pro Phe Val Ser Asp Tyr Ala Ser Tyr Ser Lys Lys Ala Ile Ser Lys
610 615 620
Leu Leu Pro Ile Met Arg Met Gly Lys Tyr Trp Ser Glu Ser Ala Val
625 630 635 640
Pro Thr Gln Val Lys Glu Arg Ser Leu Ser Ile Met Glu Arg Val Lys
645 650 655
Val Leu Pro Leu Lys Glu Gly Tyr Ser Asp Lys Asp Leu Ala Asp Leu
660 665 670
Leu Ser Arg Val Ser Asp Asp Asp Ile Pro Lys Gln Leu Ile Lys Ser
675 680 685
Phe Ile Ser Phe Lys Asp Lys Asn Pro Leu Lys Gly Leu Asn Thr Tyr
690 695 700
Gln Ala Asn Tyr Leu Val Tyr Gly Arg His Ser Glu Thr Gly Asp Ile
705 710 715 720
Gln His Trp Lys Thr Pro Glu Asp Ile Asp Arg Tyr Leu Asn Asn Phe
725 730 735
Lys Gln His Ser Leu Arg Asn Pro Ile Val Glu Gln Val Val Met Glu
740 745 750
Thr Leu Arg Val Val Arg Asp Ile Trp Glu His Tyr Gly Asn Asn Glu
755 760 765
Lys Asp Phe Phe Lys Glu Ile His Val Glu Leu Gly Arg Glu Met Lys
770 775 780
Ser Pro Ala Gly Lys Arg Glu Lys Leu Ser Gln Arg Asn Thr Glu Asn
785 790 795 800
Glu Asn Thr Asn His Arg Ile Arg Glu Val Leu Lys Glu Leu Met Asn
805 810 815
Asp Ala Ser Val Glu Gly Gly Val Arg Asp Tyr Ser Pro Ser Gln Gln
820 825 830
Glu Ile Leu Lys Leu Tyr Glu Glu Gly Ile Tyr Gln Asn Pro Asn Thr
835 840 845
Asn Tyr Leu Lys Val Asp Glu Asp Glu Ile Leu Lys Ile Arg Lys Lys
850 855 860
Asn Asn Pro Thr Gln Lys Glu Ile Gln Arg Tyr Lys Leu Trp Leu Glu
865 870 875 880
Gln Gly Tyr Ile Ser Pro Tyr Thr Gly Lys Ile Ile Pro Leu Thr Lys
885 890 895
Leu Phe Thr His Glu Tyr Gln Ile Glu His Ile Ile Pro Gln Ser Arg
900 905 910
Tyr Tyr Asp Asn Ser Leu Gly Asn Lys Ile Ile Cys Glu Ser Glu Val
915 920 925
Asn Glu Asp Lys Asp Asn Lys Thr Ala Tyr Glu Tyr Leu Lys Val Glu
930 935 940
Lys Gly Ser Ile Val Phe Gly His Lys Leu Leu Asn Leu Asp Glu Tyr
945 950 955 960
Glu Ala His Val Asn Lys Tyr Phe Lys Lys Asn Lys Thr Lys Leu Lys
965 970 975
Asn Leu Leu Ser Glu Asp Ile Pro Glu Gly Phe Ile Asn Arg Gln Leu
980 985 990
Asn Asp Ser Arg Tyr Ile Ser Lys Leu Val Lys Gly Leu Leu Ser Asn
995 1000 1005
Ile Val Arg Glu Asn Gly Glu Gln Glu Ala Thr Ser Lys Asn Leu
1010 1015 1020
Ile Pro Val Thr Gly Val Val Thr Ser Lys Leu Lys Gln Asp Trp
1025 1030 1035
Gly Leu Asn Asp Lys Trp Asn Glu Ile Ile Ala Pro Arg Phe Lys
1040 1045 1050
Arg Leu Asn Lys Leu Thr Asn Ser Asn Asp Phe Gly Phe Trp Asp
1055 1060 1065
Asn Asp Ile Asn Ala Phe Arg Ile Gln Val Pro Asp Ser Leu Ile
1070 1075 1080
Lys Gly Phe Ser Lys Lys Arg Ile Asp His Arg His His Ala Leu
1085 1090 1095
Asp Ala Leu Val Val Ala Cys Thr Ser Arg Asn His Thr His Tyr
1100 1105 1110
Leu Ser Ala Leu Asn Ala Glu Asn Lys Asn Tyr Ser Leu Arg Asp
1115 1120 1125
Lys Leu Val Ile Lys Asn Glu Asn Gly Asp Tyr Thr Lys Thr Phe
1130 1135 1140
Gln Ile Pro Trp Gln Gly Phe Thr Ile Glu Ala Lys Asn Asn Leu
1145 1150 1155
Glu Lys Thr Val Val Ser Phe Lys Lys Asn Leu Arg Val Ile Asn
1160 1165 1170
Lys Thr Asn Asn Lys Phe Trp Ser Tyr Lys Asp Glu Asn Gly Asn
1175 1180 1185
Leu Asn Leu Gly Lys Asp Gly Lys Pro Lys Lys Lys Leu Arg Lys
1190 1195 1200
Gln Thr Lys Gly Tyr Asn Trp Ala Ile Arg Lys Pro Leu His Lys
1205 1210 1215
Glu Thr Val Ser Gly Ile Tyr Asn Ile Asn Ala Pro Lys Asn Lys
1220 1225 1230
Ile Ala Thr Ser Val Arg Thr Leu Leu Thr Glu Ile Lys Asn Glu
1235 1240 1245
Lys His Leu Ala Lys Ile Thr Asp Leu Arg Ile Arg Glu Thr Ile
1250 1255 1260
Leu Pro Asn His Leu Lys His Tyr Leu Asn Asn Lys Gly Glu Ala
1265 1270 1275
Asn Phe Ser Glu Ala Phe Ser Gln Gly Gly Ile Glu Asp Leu Asn
1280 1285 1290
Lys Lys Ile Thr Thr Leu Asn Glu Gly Lys Lys His Gln Pro Ile
1295 1300 1305
Tyr Arg Val Lys Ile Phe Glu Val Gly Ser Lys Phe Ser Ile Ser
1310 1315 1320
Glu Asp Glu Asn Ser Ala Lys Ser Lys Lys Tyr Val Glu Ala Ala
1325 1330 1335
Lys Gly Thr Asn Leu Phe Phe Ala Ile Tyr Leu Asp Glu Glu Asn
1340 1345 1350
Lys Lys Arg Asn Tyr Glu Thr Ile Pro Leu Asn Glu Val Ile Thr
1355 1360 1365
His Gln Lys Gln Val Ala Gly Phe Pro Lys Ser Glu Arg Leu Ser
1370 1375 1380
Val Gln Pro Asp Ser Gln Lys Gly Thr Phe Leu Phe Thr Leu Ser
1385 1390 1395
Pro Asn Asp Leu Val Tyr Val Pro Asn Asn Glu Glu Leu Glu Asn
1400 1405 1410
Arg Asp Leu Phe Asn Leu Gly Asn Leu Asn Val Glu Gln Ile Ser
1415 1420 1425
Arg Ile Tyr Lys Phe Thr Asp Ser Ser Asp Lys Thr Cys Asn Phe
1430 1435 1440
Ile Pro Phe Gln Val Ser Lys Leu Ile Phe Asn Leu Lys Lys Lys
1445 1450 1455
Glu Gln Lys Lys Leu Asp Val Asp Phe Ile Ile Gln Asn Glu Phe
1460 1465 1470
Gly Leu Gly Ser Pro Gln Ser Lys Asn Gln Lys Ser Ile Asp Asp
1475 1480 1485
Val Met Ile Lys Glu Lys Cys Ile Lys Leu Lys Ile Asp Arg Leu
1490 1495 1500
Gly Asn Ile Ser Lys Ala
1505
<210> 27
<211> 1388
<212> PRT
<213> Artificial Sequence
<220>
<223> Cas9 from Streptococcus thermophilus
<400> 27
Met Thr Lys Pro Tyr Ser Ile Gly Leu Asp Ile Gly Thr Asn Ser Val
1 5 10 15
Gly Trp Ala Val Thr Thr Asp Asn Tyr Lys Val Pro Ser Lys Lys Met
20 25 30
Lys Val Leu Gly Asn Thr Ser Lys Lys Tyr Ile Lys Lys Asn Leu Leu
35 40 45
Gly Val Leu Leu Phe Asp Ser Gly Ile Thr Ala Glu Gly Arg Arg Leu
50 55 60
Lys Arg Thr Ala Arg Arg Arg Tyr Thr Arg Arg Arg Asn Arg Ile Leu
65 70 75 80
Tyr Leu Gln Glu Ile Phe Ser Thr Glu Met Ala Thr Leu Asp Asp Ala
85 90 95
Phe Phe Gln Arg Leu Asp Asp Ser Phe Leu Val Pro Asp Asp Lys Arg
100 105 110
Asp Ser Lys Tyr Pro Ile Phe Gly Asn Leu Val Glu Glu Lys Ala Tyr
115 120 125
His Asp Glu Phe Pro Thr Ile Tyr His Leu Arg Lys Tyr Leu Ala Asp
130 135 140
Ser Thr Lys Lys Ala Asp Leu Arg Leu Val Tyr Leu Ala Leu Ala His
145 150 155 160
Met Ile Lys Tyr Arg Gly His Phe Leu Ile Glu Gly Glu Phe Asn Ser
165 170 175
Lys Asn Asn Asp Ile Gln Lys Asn Phe Gln Asp Phe Leu Asp Thr Tyr
180 185 190
Asn Ala Ile Phe Glu Ser Asp Leu Ser Leu Glu Asn Ser Lys Gln Leu
195 200 205
Glu Glu Ile Val Lys Asp Lys Ile Ser Lys Leu Glu Lys Lys Asp Arg
210 215 220
Ile Leu Lys Leu Phe Pro Gly Glu Lys Asn Ser Gly Ile Phe Ser Glu
225 230 235 240
Phe Leu Lys Leu Ile Val Gly Asn Gln Ala Asp Phe Arg Lys Cys Phe
245 250 255
Asn Leu Asp Glu Lys Ala Ser Leu His Phe Ser Lys Glu Ser Tyr Asp
260 265 270
Glu Asp Leu Glu Thr Leu Leu Gly Tyr Ile Gly Asp Asp Tyr Ser Asp
275 280 285
Val Phe Leu Lys Ala Lys Lys Leu Tyr Asp Ala Ile Leu Leu Ser Gly
290 295 300
Phe Leu Thr Val Thr Asp Asn Glu Thr Glu Ala Pro Leu Ser Ser Ala
305 310 315 320
Met Ile Lys Arg Tyr Asn Glu His Lys Glu Asp Leu Ala Leu Leu Lys
325 330 335
Glu Tyr Ile Arg Asn Ile Ser Leu Lys Thr Tyr Asn Glu Val Phe Lys
340 345 350
Asp Asp Thr Lys Asn Gly Tyr Ala Gly Tyr Ile Asp Gly Lys Thr Asn
355 360 365
Gln Glu Asp Phe Tyr Val Tyr Leu Lys Lys Leu Leu Ala Glu Phe Glu
370 375 380
Gly Ala Asp Tyr Phe Leu Glu Lys Ile Asp Arg Glu Asp Phe Leu Arg
385 390 395 400
Lys Gln Arg Thr Phe Asp Asn Gly Ser Ile Pro Tyr Gln Ile His Leu
405 410 415
Gln Glu Met Arg Ala Ile Leu Asp Lys Gln Ala Lys Phe Tyr Pro Phe
420 425 430
Leu Ala Lys Asn Lys Glu Arg Ile Glu Lys Ile Leu Thr Phe Arg Ile
435 440 445
Pro Tyr Tyr Val Gly Pro Leu Ala Arg Gly Asn Ser Asp Phe Ala Trp
450 455 460
Ser Ile Arg Lys Arg Asn Glu Lys Ile Thr Pro Trp Asn Phe Glu Asp
465 470 475 480
Val Ile Asp Lys Glu Ser Ser Ala Glu Ala Phe Ile Asn Arg Met Thr
485 490 495
Ser Phe Asp Leu Tyr Leu Pro Glu Glu Lys Val Leu Pro Lys His Ser
500 505 510
Leu Leu Tyr Glu Thr Phe Asn Val Tyr Asn Glu Leu Thr Lys Val Arg
515 520 525
Phe Ile Ala Glu Ser Met Arg Asp Tyr Gln Phe Leu Asp Ser Lys Gln
530 535 540
Lys Lys Asp Ile Val Arg Leu Tyr Phe Lys Asp Lys Arg Lys Val Thr
545 550 555 560
Asp Lys Asp Ile Ile Glu Tyr Leu His Ala Ile Tyr Gly Tyr Asp Gly
565 570 575
Ile Glu Leu Lys Gly Ile Glu Lys Gln Phe Asn Ser Ser Leu Ser Thr
580 585 590
Tyr His Asp Leu Leu Asn Ile Ile Asn Asp Lys Glu Phe Leu Asp Asp
595 600 605
Ser Ser Asn Glu Ala Ile Ile Glu Glu Ile Ile His Thr Leu Thr Ile
610 615 620
Phe Glu Asp Arg Glu Met Ile Lys Gln Arg Leu Ser Lys Phe Glu Asn
625 630 635 640
Ile Phe Asp Lys Ser Val Leu Lys Lys Leu Ser Arg Arg His Tyr Thr
645 650 655
Gly Trp Gly Lys Leu Ser Ala Lys Leu Ile Asn Gly Ile Arg Asp Glu
660 665 670
Lys Ser Gly Asn Thr Ile Leu Asp Tyr Leu Ile Asp Asp Gly Ile Ser
675 680 685
Asn Arg Asn Phe Met Gln Leu Ile His Asp Asp Ala Leu Ser Phe Lys
690 695 700
Lys Lys Ile Gln Lys Ala Gln Ile Ile Gly Asp Glu Asp Lys Gly Asn
705 710 715 720
Ile Lys Glu Val Val Lys Ser Leu Pro Gly Ser Pro Ala Ile Lys Lys
725 730 735
Gly Ile Leu Gln Ser Ile Lys Ile Val Asp Glu Leu Val Lys Val Met
740 745 750
Gly Gly Arg Lys Pro Glu Ser Ile Val Val Glu Met Ala Arg Glu Asn
755 760 765
Gln Tyr Thr Asn Gln Gly Lys Ser Asn Ser Gln Gln Arg Leu Lys Arg
770 775 780
Leu Glu Lys Ser Leu Lys Glu Leu Gly Ser Lys Ile Leu Lys Glu Asn
785 790 795 800
Ile Pro Ala Lys Leu Ser Lys Ile Asp Asn Asn Ala Leu Gln Asn Asp
805 810 815
Arg Leu Tyr Leu Tyr Tyr Leu Gln Asn Gly Lys Asp Met Tyr Thr Gly
820 825 830
Asp Asp Leu Asp Ile Asp Arg Leu Ser Asn Tyr Asp Ile Asp His Ile
835 840 845
Ile Pro Gln Ala Phe Leu Lys Asp Asn Ser Ile Asp Asn Lys Val Leu
850 855 860
Val Ser Ser Ala Ser Asn Arg Gly Lys Ser Asp Asp Val Pro Ser Leu
865 870 875 880
Glu Val Val Lys Lys Arg Lys Thr Phe Trp Tyr Gln Leu Leu Lys Ser
885 890 895
Lys Leu Ile Ser Gln Arg Lys Phe Asp Asn Leu Thr Lys Ala Glu Arg
900 905 910
Gly Gly Leu Ser Pro Glu Asp Lys Ala Gly Phe Ile Gln Arg Gln Leu
915 920 925
Val Glu Thr Arg Gln Ile Thr Lys His Val Ala Arg Leu Leu Asp Glu
930 935 940
Lys Phe Asn Asn Lys Lys Asp Glu Asn Asn Arg Ala Val Arg Thr Val
945 950 955 960
Lys Ile Ile Thr Leu Lys Ser Thr Leu Val Ser Gln Phe Arg Lys Asp
965 970 975
Phe Glu Leu Tyr Lys Val Arg Glu Ile Asn Asp Phe His His Ala His
980 985 990
Asp Ala Tyr Leu Asn Ala Val Val Ala Ser Ala Leu Leu Lys Lys Tyr
995 1000 1005
Pro Lys Leu Glu Pro Glu Phe Val Tyr Gly Asp Tyr Pro Lys Tyr
1010 1015 1020
Asn Ser Phe Arg Glu Arg Lys Ser Ala Thr Glu Lys Val Tyr Phe
1025 1030 1035
Tyr Ser Asn Ile Met Asn Ile Phe Lys Lys Ser Ile Ser Leu Ala
1040 1045 1050
Asp Gly Arg Val Ile Glu Arg Pro Leu Ile Glu Val Asn Glu Glu
1055 1060 1065
Thr Gly Glu Ser Val Trp Asn Lys Glu Ser Asp Leu Ala Thr Val
1070 1075 1080
Arg Arg Val Leu Ser Tyr Pro Gln Val Asn Val Val Lys Lys Val
1085 1090 1095
Glu Glu Gln Asn His Gly Leu Asp Arg Gly Lys Pro Lys Gly Leu
1100 1105 1110
Phe Asn Ala Asn Leu Ser Ser Lys Pro Lys Pro Asn Ser Asn Glu
1115 1120 1125
Asn Leu Val Gly Ala Lys Glu Tyr Leu Asp Pro Lys Lys Tyr Gly
1130 1135 1140
Gly Tyr Ala Gly Ile Ser Asn Ser Phe Thr Val Leu Val Lys Gly
1145 1150 1155
Thr Ile Glu Lys Gly Ala Lys Lys Lys Ile Thr Asn Val Leu Glu
1160 1165 1170
Phe Gln Gly Ile Ser Ile Leu Asp Arg Ile Asn Tyr Arg Lys Asp
1175 1180 1185
Lys Leu Asn Phe Leu Leu Glu Lys Gly Tyr Lys Asp Ile Glu Leu
1190 1195 1200
Ile Ile Glu Leu Pro Lys Tyr Ser Leu Phe Glu Leu Ser Asp Gly
1205 1210 1215
Ser Arg Arg Met Leu Ala Ser Ile Leu Ser Thr Asn Asn Lys Arg
1220 1225 1230
Gly Glu Ile His Lys Gly Asn Gln Ile Phe Leu Ser Gln Lys Phe
1235 1240 1245
Val Lys Leu Leu Tyr His Ala Lys Arg Ile Ser Asn Thr Ile Asn
1250 1255 1260
Glu Asn His Arg Lys Tyr Val Glu Asn His Lys Lys Glu Phe Glu
1265 1270 1275
Glu Leu Phe Tyr Tyr Ile Leu Glu Phe Asn Glu Asn Tyr Val Gly
1280 1285 1290
Ala Lys Lys Asn Gly Lys Leu Leu Asn Ser Ala Phe Gln Ser Trp
1295 1300 1305
Gln Asn His Ser Ile Asp Glu Leu Cys Ser Ser Phe Ile Gly Pro
1310 1315 1320
Thr Gly Ser Glu Arg Lys Gly Leu Phe Glu Leu Thr Ser Arg Gly
1325 1330 1335
Ser Ala Ala Asp Phe Glu Phe Leu Gly Val Lys Ile Pro Arg Tyr
1340 1345 1350
Arg Asp Tyr Thr Pro Ser Ser Leu Leu Lys Asp Ala Thr Leu Ile
1355 1360 1365
His Gln Ser Val Thr Gly Leu Tyr Glu Thr Arg Ile Asp Leu Ala
1370 1375 1380
Lys Leu Gly Glu Gly
1385
<210> 28
<211> 1334
<212> PRT
<213> Artificial Sequence
<220>
<223> Cas9 from Listeria innocua
<400> 28
Met Lys Lys Pro Tyr Thr Ile Gly Leu Asp Ile Gly Thr Asn Ser Val
1 5 10 15
Gly Trp Ala Val Leu Thr Asp Gln Tyr Asp Leu Val Lys Arg Lys Met
20 25 30
Lys Ile Ala Gly Asp Ser Glu Lys Lys Gln Ile Lys Lys Asn Phe Trp
35 40 45
Gly Val Arg Leu Phe Asp Glu Gly Gln Thr Ala Ala Asp Arg Arg Met
50 55 60
Ala Arg Thr Ala Arg Arg Arg Ile Glu Arg Arg Arg Asn Arg Ile Ser
65 70 75 80
Tyr Leu Gln Gly Ile Phe Ala Glu Glu Met Ser Lys Thr Asp Ala Asn
85 90 95
Phe Phe Cys Arg Leu Ser Asp Ser Phe Tyr Val Asp Asn Glu Lys Arg
100 105 110
Asn Ser Arg His Pro Phe Phe Ala Thr Ile Glu Glu Glu Val Glu Tyr
115 120 125
His Lys Asn Tyr Pro Thr Ile Tyr His Leu Arg Glu Glu Leu Val Asn
130 135 140
Ser Ser Glu Lys Ala Asp Leu Arg Leu Val Tyr Leu Ala Leu Ala His
145 150 155 160
Ile Ile Lys Tyr Arg Gly Asn Phe Leu Ile Glu Gly Ala Leu Asp Thr
165 170 175
Gln Asn Thr Ser Val Asp Gly Ile Tyr Lys Gln Phe Ile Gln Thr Tyr
180 185 190
Asn Gln Val Phe Ala Ser Gly Ile Glu Asp Gly Ser Leu Lys Lys Leu
195 200 205
Glu Asp Asn Lys Asp Val Ala Lys Ile Leu Val Glu Lys Val Thr Arg
210 215 220
Lys Glu Lys Leu Glu Arg Ile Leu Lys Leu Tyr Pro Gly Glu Lys Ser
225 230 235 240
Ala Gly Met Phe Ala Gln Phe Ile Ser Leu Ile Val Gly Ser Lys Gly
245 250 255
Asn Phe Gln Lys Pro Phe Asp Leu Ile Glu Lys Ser Asp Ile Glu Cys
260 265 270
Ala Lys Asp Ser Tyr Glu Glu Asp Leu Glu Ser Leu Leu Ala Leu Ile
275 280 285
Gly Asp Glu Tyr Ala Glu Leu Phe Val Ala Ala Lys Asn Ala Tyr Ser
290 295 300
Ala Val Val Leu Ser Ser Ile Ile Thr Val Ala Glu Thr Glu Thr Asn
305 310 315 320
Ala Lys Leu Ser Ala Ser Met Ile Glu Arg Phe Asp Thr His Glu Glu
325 330 335
Asp Leu Gly Glu Leu Lys Ala Phe Ile Lys Leu His Leu Pro Lys His
340 345 350
Tyr Glu Glu Ile Phe Ser Asn Thr Glu Lys His Gly Tyr Ala Gly Tyr
355 360 365
Ile Asp Gly Lys Thr Lys Gln Ala Asp Phe Tyr Lys Tyr Met Lys Met
370 375 380
Thr Leu Glu Asn Ile Glu Gly Ala Asp Tyr Phe Ile Ala Lys Ile Glu
385 390 395 400
Lys Glu Asn Phe Leu Arg Lys Gln Arg Thr Phe Asp Asn Gly Ala Ile
405 410 415
Pro His Gln Leu His Leu Glu Glu Leu Glu Ala Ile Leu His Gln Gln
420 425 430
Ala Lys Tyr Tyr Pro Phe Leu Lys Glu Asn Tyr Asp Lys Ile Lys Ser
435 440 445
Leu Val Thr Phe Arg Ile Pro Tyr Phe Val Gly Pro Leu Ala Asn Gly
450 455 460
Gln Ser Glu Phe Ala Trp Leu Thr Arg Lys Ala Asp Gly Glu Ile Arg
465 470 475 480
Pro Trp Asn Ile Glu Glu Lys Val Asp Phe Gly Lys Ser Ala Val Asp
485 490 495
Phe Ile Glu Lys Met Thr Asn Lys Asp Thr Tyr Leu Pro Lys Glu Asn
500 505 510
Val Leu Pro Lys His Ser Leu Cys Tyr Gln Lys Tyr Leu Val Tyr Asn
515 520 525
Glu Leu Thr Lys Val Arg Tyr Ile Asn Asp Gln Gly Lys Thr Ser Tyr
530 535 540
Phe Ser Gly Gln Glu Lys Glu Gln Ile Phe Asn Asp Leu Phe Lys Gln
545 550 555 560
Lys Arg Lys Val Lys Lys Lys Asp Leu Glu Leu Phe Leu Arg Asn Met
565 570 575
Ser His Val Glu Ser Pro Thr Ile Glu Gly Leu Glu Asp Ser Phe Asn
580 585 590
Ser Ser Tyr Ser Thr Tyr His Asp Leu Leu Lys Val Gly Ile Lys Gln
595 600 605
Glu Ile Leu Asp Asn Pro Val Asn Thr Glu Met Leu Glu Asn Ile Val
610 615 620
Lys Ile Leu Thr Val Phe Glu Asp Lys Arg Met Ile Lys Glu Gln Leu
625 630 635 640
Gln Gln Phe Ser Asp Val Leu Asp Gly Val Val Leu Lys Lys Leu Glu
645 650 655
Arg Arg His Tyr Thr Gly Trp Gly Arg Leu Ser Ala Lys Leu Leu Met
660 665 670
Gly Ile Arg Asp Lys Gln Ser His Leu Thr Ile Leu Asp Tyr Leu Met
675 680 685
Asn Asp Asp Gly Leu Asn Arg Asn Leu Met Gln Leu Ile Asn Asp Ser
690 695 700
Asn Leu Ser Phe Lys Ser Ile Ile Glu Lys Glu Gln Val Thr Thr Ala
705 710 715 720
Asp Lys Asp Ile Gln Ser Ile Val Ala Asp Leu Ala Gly Ser Pro Ala
725 730 735
Ile Lys Lys Gly Ile Leu Gln Ser Leu Lys Ile Val Asp Glu Leu Val
740 745 750
Ser Val Met Gly Tyr Pro Pro Gln Thr Ile Val Val Glu Met Ala Arg
755 760 765
Glu Asn Gln Thr Thr Gly Lys Gly Lys Asn Asn Ser Arg Pro Arg Tyr
770 775 780
Lys Ser Leu Glu Lys Ala Ile Lys Glu Phe Gly Ser Gln Ile Leu Lys
785 790 795 800
Glu His Pro Thr Asp Asn Gln Glu Leu Arg Asn Asn Arg Leu Tyr Leu
805 810 815
Tyr Tyr Leu Gln Asn Gly Lys Asp Met Tyr Thr Gly Gln Asp Leu Asp
820 825 830
Ile His Asn Leu Ser Asn Tyr Asp Ile Asp His Ile Val Pro Gln Ser
835 840 845
Phe Ile Thr Asp Asn Ser Ile Asp Asn Leu Val Leu Thr Ser Ser Ala
850 855 860
Gly Asn Arg Glu Lys Gly Asp Asp Val Pro Pro Leu Glu Ile Val Arg
865 870 875 880
Lys Arg Lys Val Phe Trp Glu Lys Leu Tyr Gln Gly Asn Leu Met Ser
885 890 895
Lys Arg Lys Phe Asp Tyr Leu Thr Lys Ala Glu Arg Gly Gly Leu Thr
900 905 910
Glu Ala Asp Lys Ala Arg Phe Ile His Arg Gln Leu Val Glu Thr Arg
915 920 925
Gln Ile Thr Lys Asn Val Ala Asn Ile Leu His Gln Arg Phe Asn Tyr
930 935 940
Glu Lys Asp Asp His Gly Asn Thr Met Lys Gln Val Arg Ile Val Thr
945 950 955 960
Leu Lys Ser Ala Leu Val Ser Gln Phe Arg Lys Gln Phe Gln Leu Tyr
965 970 975
Lys Val Arg Asp Val Asn Asp Tyr His His Ala His Asp Ala Tyr Leu
980 985 990
Asn Gly Val Val Ala Asn Thr Leu Leu Lys Val Tyr Pro Gln Leu Glu
995 1000 1005
Pro Glu Phe Val Tyr Gly Asp Tyr His Gln Phe Asp Trp Phe Lys
1010 1015 1020
Ala Asn Lys Ala Thr Ala Lys Lys Gln Phe Tyr Thr Asn Ile Met
1025 1030 1035
Leu Phe Phe Ala Gln Lys Asp Arg Ile Ile Asp Glu Asn Gly Glu
1040 1045 1050
Ile Leu Trp Asp Lys Lys Tyr Leu Asp Thr Val Lys Lys Val Met
1055 1060 1065
Ser Tyr Arg Gln Met Asn Ile Val Lys Lys Thr Glu Ile Gln Lys
1070 1075 1080
Gly Glu Phe Ser Lys Ala Thr Ile Lys Pro Lys Gly Asn Ser Ser
1085 1090 1095
Lys Leu Ile Pro Arg Lys Thr Asn Trp Asp Pro Met Lys Tyr Gly
1100 1105 1110
Gly Leu Asp Ser Pro Asn Met Ala Tyr Ala Val Val Ile Glu Tyr
1115 1120 1125
Ala Lys Gly Lys Asn Lys Leu Val Phe Glu Lys Lys Ile Ile Arg
1130 1135 1140
Val Thr Ile Met Glu Arg Lys Ala Phe Glu Lys Asp Glu Lys Ala
1145 1150 1155
Phe Leu Glu Glu Gln Gly Tyr Arg Gln Pro Lys Val Leu Ala Lys
1160 1165 1170
Leu Pro Lys Tyr Thr Leu Tyr Glu Cys Glu Glu Gly Arg Arg Arg
1175 1180 1185
Met Leu Ala Ser Ala Asn Glu Ala Gln Lys Gly Asn Gln Gln Val
1190 1195 1200
Leu Pro Asn His Leu Val Thr Leu Leu His His Ala Ala Asn Cys
1205 1210 1215
Glu Val Ser Asp Gly Lys Ser Leu Asp Tyr Ile Glu Ser Asn Arg
1220 1225 1230
Glu Met Phe Ala Glu Leu Leu Ala His Val Ser Glu Phe Ala Lys
1235 1240 1245
Arg Tyr Thr Leu Ala Glu Ala Asn Leu Asn Lys Ile Asn Gln Leu
1250 1255 1260
Phe Glu Gln Asn Lys Glu Gly Asp Ile Lys Ala Ile Ala Gln Ser
1265 1270 1275
Phe Val Asp Leu Met Ala Phe Asn Ala Met Gly Ala Pro Ala Ser
1280 1285 1290
Phe Lys Phe Phe Glu Thr Thr Ile Glu Arg Lys Arg Tyr Asn Asn
1295 1300 1305
Leu Lys Glu Leu Leu Asn Ser Thr Ile Ile Tyr Gln Ser Ile Thr
1310 1315 1320
Gly Leu Tyr Glu Ser Arg Lys Arg Leu Asp Asp
1325 1330
<210> 29
<211> 984
<212> PRT
<213> Artificial Sequence
<220>
<223> Cas9 from Campylobacter jejuni
<400> 29
Met Ala Arg Ile Leu Ala Phe Asp Ile Gly Ile Ser Ser Ile Gly Trp
1 5 10 15
Ala Phe Ser Glu Asn Asp Glu Leu Lys Asp Cys Gly Val Arg Ile Phe
20 25 30
Thr Lys Val Glu Asn Pro Lys Thr Gly Glu Ser Leu Ala Leu Pro Arg
35 40 45
Arg Leu Ala Arg Ser Ala Arg Lys Arg Leu Ala Arg Arg Lys Ala Arg
50 55 60
Leu Asn His Leu Lys His Leu Ile Ala Asn Glu Phe Lys Leu Asn Tyr
65 70 75 80
Glu Asp Tyr Gln Ser Phe Asp Glu Ser Leu Ala Lys Ala Tyr Lys Gly
85 90 95
Ser Leu Ile Ser Pro Tyr Glu Leu Arg Phe Arg Ala Leu Asn Glu Leu
100 105 110
Leu Ser Lys Gln Asp Phe Ala Arg Val Ile Leu His Ile Ala Lys Arg
115 120 125
Arg Gly Tyr Asp Asp Ile Lys Asn Ser Asp Asp Lys Glu Lys Gly Ala
130 135 140
Ile Leu Lys Ala Ile Lys Gln Asn Glu Glu Lys Leu Ala Asn Tyr Gln
145 150 155 160
Ser Val Gly Glu Tyr Leu Tyr Lys Glu Tyr Phe Gln Lys Phe Lys Glu
165 170 175
Asn Ser Lys Glu Phe Thr Asn Val Arg Asn Lys Lys Glu Ser Tyr Glu
180 185 190
Arg Cys Ile Ala Gln Ser Phe Leu Lys Asp Glu Leu Lys Leu Ile Phe
195 200 205
Lys Lys Gln Arg Glu Phe Gly Phe Ser Phe Ser Lys Lys Phe Glu Glu
210 215 220
Glu Val Leu Ser Val Ala Phe Tyr Lys Arg Ala Leu Lys Asp Phe Ser
225 230 235 240
His Leu Val Gly Asn Cys Ser Phe Phe Thr Asp Glu Lys Arg Ala Pro
245 250 255
Lys Asn Ser Pro Leu Ala Phe Met Phe Val Ala Leu Thr Arg Ile Ile
260 265 270
Asn Leu Leu Asn Asn Leu Lys Asn Thr Glu Gly Ile Leu Tyr Thr Lys
275 280 285
Asp Asp Leu Asn Ala Leu Leu Asn Glu Val Leu Lys Asn Gly Thr Leu
290 295 300
Thr Tyr Lys Gln Thr Lys Lys Leu Leu Gly Leu Ser Asp Asp Tyr Glu
305 310 315 320
Phe Lys Gly Glu Lys Gly Thr Tyr Phe Ile Glu Phe Lys Lys Tyr Lys
325 330 335
Glu Phe Ile Lys Ala Leu Gly Glu His Asn Leu Ser Gln Asp Asp Leu
340 345 350
Asn Glu Ile Ala Lys Asp Ile Thr Leu Ile Lys Asp Glu Ile Lys Leu
355 360 365
Lys Lys Ala Leu Ala Lys Tyr Asp Leu Asn Gln Asn Gln Ile Asp Ser
370 375 380
Leu Ser Lys Leu Glu Phe Lys Asp His Leu Asn Ile Ser Phe Lys Ala
385 390 395 400
Leu Lys Leu Val Thr Pro Leu Met Leu Glu Gly Lys Lys Tyr Asp Glu
405 410 415
Ala Cys Asn Glu Leu Asn Leu Lys Val Ala Ile Asn Glu Asp Lys Lys
420 425 430
Asp Phe Leu Pro Ala Phe Asn Glu Thr Tyr Tyr Lys Asp Glu Val Thr
435 440 445
Asn Pro Val Val Leu Arg Ala Ile Lys Glu Tyr Arg Lys Val Leu Asn
450 455 460
Ala Leu Leu Lys Lys Tyr Gly Lys Val His Lys Ile Asn Ile Glu Leu
465 470 475 480
Ala Arg Glu Val Gly Lys Asn His Ser Gln Arg Ala Lys Ile Glu Lys
485 490 495
Glu Gln Asn Glu Asn Tyr Lys Ala Lys Lys Asp Ala Glu Leu Glu Cys
500 505 510
Glu Lys Leu Gly Leu Lys Ile Asn Ser Lys Asn Ile Leu Lys Leu Arg
515 520 525
Leu Phe Lys Glu Gln Lys Glu Phe Cys Ala Tyr Ser Gly Glu Lys Ile
530 535 540
Lys Ile Ser Asp Leu Gln Asp Glu Lys Met Leu Glu Ile Asp His Ile
545 550 555 560
Tyr Pro Tyr Ser Arg Ser Phe Asp Asp Ser Tyr Met Asn Lys Val Leu
565 570 575
Val Phe Thr Lys Gln Asn Gln Glu Lys Leu Asn Gln Thr Pro Phe Glu
580 585 590
Ala Phe Gly Asn Asp Ser Ala Lys Trp Gln Lys Ile Glu Val Leu Ala
595 600 605
Lys Asn Leu Pro Thr Lys Lys Gln Lys Arg Ile Leu Asp Lys Asn Tyr
610 615 620
Lys Asp Lys Glu Gln Lys Asn Phe Lys Asp Arg Asn Leu Asn Asp Thr
625 630 635 640
Arg Tyr Ile Ala Arg Leu Val Leu Asn Tyr Thr Lys Asp Tyr Leu Asp
645 650 655
Phe Leu Pro Leu Ser Asp Asp Glu Asn Thr Lys Leu Asn Asp Thr Gln
660 665 670
Lys Gly Ser Lys Val His Val Glu Ala Lys Ser Gly Met Leu Thr Ser
675 680 685
Ala Leu Arg His Thr Trp Gly Phe Ser Ala Lys Asp Arg Asn Asn His
690 695 700
Leu His His Ala Ile Asp Ala Val Ile Ile Ala Tyr Ala Asn Asn Ser
705 710 715 720
Ile Val Lys Ala Phe Ser Asp Phe Lys Lys Glu Gln Glu Ser Asn Ser
725 730 735
Ala Glu Leu Tyr Ala Lys Lys Ile Ser Glu Leu Asp Tyr Lys Asn Lys
740 745 750
Arg Lys Phe Phe Glu Pro Phe Ser Gly Phe Arg Gln Lys Val Leu Asp
755 760 765
Lys Ile Asp Glu Ile Phe Val Ser Lys Pro Glu Arg Lys Lys Pro Ser
770 775 780
Gly Ala Leu His Glu Glu Thr Phe Arg Lys Glu Glu Glu Phe Tyr Gln
785 790 795 800
Ser Tyr Gly Gly Lys Glu Gly Val Leu Lys Ala Leu Glu Leu Gly Lys
805 810 815
Ile Arg Lys Val Asn Gly Lys Ile Val Lys Asn Gly Asp Met Phe Arg
820 825 830
Val Asp Ile Phe Lys His Lys Lys Thr Asn Lys Phe Tyr Ala Val Pro
835 840 845
Ile Tyr Thr Met Asp Phe Ala Leu Lys Val Leu Pro Asn Lys Ala Val
850 855 860
Ala Arg Ser Lys Lys Gly Glu Ile Lys Asp Trp Ile Leu Met Asp Glu
865 870 875 880
Asn Tyr Glu Phe Cys Phe Ser Leu Tyr Lys Asp Ser Leu Ile Leu Ile
885 890 895
Gln Thr Lys Asp Met Gln Glu Pro Glu Phe Val Tyr Tyr Asn Ala Phe
900 905 910
Thr Ser Ser Thr Val Ser Leu Ile Val Ser Lys His Asp Asn Lys Phe
915 920 925
Glu Thr Leu Ser Lys Asn Gln Lys Ile Leu Phe Lys Asn Ala Asn Glu
930 935 940
Lys Glu Val Ile Ala Lys Ser Ile Gly Ile Gln Asn Leu Lys Val Phe
945 950 955 960
Glu Lys Tyr Ile Val Ser Ala Leu Gly Glu Val Thr Lys Ala Glu Phe
965 970 975
Arg Gln Arg Glu Asp Phe Lys Lys
980
<210> 30
<211> 1082
<212> PRT
<213> Artificial Sequence
<220>
<223> Cas9 from Neisseria. meningitidis
<400> 30
Met Ala Ala Phe Lys Pro Asn Pro Ile Asn Tyr Ile Leu Gly Leu Asp
1 5 10 15
Ile Gly Ile Ala Ser Val Gly Trp Ala Met Val Glu Ile Asp Glu Asp
20 25 30
Glu Asn Pro Ile Cys Leu Ile Asp Leu Gly Val Arg Val Phe Glu Arg
35 40 45
Ala Glu Val Pro Lys Thr Gly Asp Ser Leu Ala Met Ala Arg Arg Leu
50 55 60
Ala Arg Ser Val Arg Arg Leu Thr Arg Arg Arg Ala His Arg Leu Leu
65 70 75 80
Arg Ala Arg Arg Leu Leu Lys Arg Glu Gly Val Leu Gln Ala Ala Asp
85 90 95
Phe Asp Glu Asn Gly Leu Ile Lys Ser Leu Pro Asn Thr Pro Trp Gln
100 105 110
Leu Arg Ala Ala Ala Leu Asp Arg Lys Leu Thr Pro Leu Glu Trp Ser
115 120 125
Ala Val Leu Leu His Leu Ile Lys His Arg Gly Tyr Leu Ser Gln Arg
130 135 140
Lys Asn Glu Gly Glu Thr Ala Asp Lys Glu Leu Gly Ala Leu Leu Lys
145 150 155 160
Gly Val Ala Asp Asn Ala His Ala Leu Gln Thr Gly Asp Phe Arg Thr
165 170 175
Pro Ala Glu Leu Ala Leu Asn Lys Phe Glu Lys Glu Ser Gly His Ile
180 185 190
Arg Asn Gln Arg Gly Asp Tyr Ser His Thr Phe Ser Arg Lys Asp Leu
195 200 205
Gln Ala Glu Leu Ile Leu Leu Phe Glu Lys Gln Lys Glu Phe Gly Asn
210 215 220
Pro His Val Ser Gly Gly Leu Lys Glu Gly Ile Glu Thr Leu Leu Met
225 230 235 240
Thr Gln Arg Pro Ala Leu Ser Gly Asp Ala Val Gln Lys Met Leu Gly
245 250 255
His Cys Thr Phe Glu Pro Ala Glu Pro Lys Ala Ala Lys Asn Thr Tyr
260 265 270
Thr Ala Glu Arg Phe Ile Trp Leu Thr Lys Leu Asn Asn Leu Arg Ile
275 280 285
Leu Glu Gln Gly Ser Glu Arg Pro Leu Thr Asp Thr Glu Arg Ala Thr
290 295 300
Leu Met Asp Glu Pro Tyr Arg Lys Ser Lys Leu Thr Tyr Ala Gln Ala
305 310 315 320
Arg Lys Leu Leu Gly Leu Glu Asp Thr Ala Phe Phe Lys Gly Leu Arg
325 330 335
Tyr Gly Lys Asp Asn Ala Glu Ala Ser Thr Leu Met Glu Met Lys Ala
340 345 350
Tyr His Ala Ile Ser Arg Ala Leu Glu Lys Glu Gly Leu Lys Asp Lys
355 360 365
Lys Ser Pro Leu Asn Leu Ser Pro Glu Leu Gln Asp Glu Ile Gly Thr
370 375 380
Ala Phe Ser Leu Phe Lys Thr Asp Glu Asp Ile Thr Gly Arg Leu Lys
385 390 395 400
Asp Arg Ile Gln Pro Glu Ile Leu Glu Ala Leu Leu Lys His Ile Ser
405 410 415
Phe Asp Lys Phe Val Gln Ile Ser Leu Lys Ala Leu Arg Arg Ile Val
420 425 430
Pro Leu Met Glu Gln Gly Lys Arg Tyr Asp Glu Ala Cys Ala Glu Ile
435 440 445
Tyr Gly Asp His Tyr Gly Lys Lys Asn Thr Glu Glu Lys Ile Tyr Leu
450 455 460
Pro Pro Ile Pro Ala Asp Glu Ile Arg Asn Pro Val Val Leu Arg Ala
465 470 475 480
Leu Ser Gln Ala Arg Lys Val Ile Asn Gly Val Val Arg Arg Tyr Gly
485 490 495
Ser Pro Ala Arg Ile His Ile Glu Thr Ala Arg Glu Val Gly Lys Ser
500 505 510
Phe Lys Asp Arg Lys Glu Ile Glu Lys Arg Gln Glu Glu Asn Arg Lys
515 520 525
Asp Arg Glu Lys Ala Ala Ala Lys Phe Arg Glu Tyr Phe Pro Asn Phe
530 535 540
Val Gly Glu Pro Lys Ser Lys Asp Ile Leu Lys Leu Arg Leu Tyr Glu
545 550 555 560
Gln Gln His Gly Lys Cys Leu Tyr Ser Gly Lys Glu Ile Asn Leu Gly
565 570 575
Arg Leu Asn Glu Lys Gly Tyr Val Glu Ile Asp His Ala Leu Pro Phe
580 585 590
Ser Arg Thr Trp Asp Asp Ser Phe Asn Asn Lys Val Leu Val Leu Gly
595 600 605
Ser Glu Asn Gln Asn Lys Gly Asn Gln Thr Pro Tyr Glu Tyr Phe Asn
610 615 620
Gly Lys Asp Asn Ser Arg Glu Trp Gln Glu Phe Lys Ala Arg Val Glu
625 630 635 640
Thr Ser Arg Phe Pro Arg Ser Lys Lys Gln Arg Ile Leu Leu Gln Lys
645 650 655
Phe Asp Glu Asp Gly Phe Lys Glu Arg Asn Leu Asn Asp Thr Arg Tyr
660 665 670
Val Asn Arg Phe Leu Cys Gln Phe Val Ala Asp Arg Met Arg Leu Thr
675 680 685
Gly Lys Gly Lys Lys Arg Val Phe Ala Ser Asn Gly Gln Ile Thr Asn
690 695 700
Leu Leu Arg Gly Phe Trp Gly Leu Arg Lys Val Arg Ala Glu Asn Asp
705 710 715 720
Arg His His Ala Leu Asp Ala Val Val Val Ala Cys Ser Thr Val Ala
725 730 735
Met Gln Gln Lys Ile Thr Arg Phe Val Arg Tyr Lys Glu Met Asn Ala
740 745 750
Phe Asp Gly Lys Thr Ile Asp Lys Glu Thr Gly Glu Val Leu His Gln
755 760 765
Lys Thr His Phe Pro Gln Pro Trp Glu Phe Phe Ala Gln Glu Val Met
770 775 780
Ile Arg Val Phe Gly Lys Pro Asp Gly Lys Pro Glu Phe Glu Glu Ala
785 790 795 800
Asp Thr Pro Glu Lys Leu Arg Thr Leu Leu Ala Glu Lys Leu Ser Ser
805 810 815
Arg Pro Glu Ala Val His Glu Tyr Val Thr Pro Leu Phe Val Ser Arg
820 825 830
Ala Pro Asn Arg Lys Met Ser Gly Gln Gly His Met Glu Thr Val Lys
835 840 845
Ser Ala Lys Arg Leu Asp Glu Gly Val Ser Val Leu Arg Val Pro Leu
850 855 860
Thr Gln Leu Lys Leu Lys Asp Leu Glu Lys Met Val Asn Arg Glu Arg
865 870 875 880
Glu Pro Lys Leu Tyr Glu Ala Leu Lys Ala Arg Leu Glu Ala His Lys
885 890 895
Asp Asp Pro Ala Lys Ala Phe Ala Glu Pro Phe Tyr Lys Tyr Asp Lys
900 905 910
Ala Gly Asn Arg Thr Gln Gln Val Lys Ala Val Arg Val Glu Gln Val
915 920 925
Gln Lys Thr Gly Val Trp Val Arg Asn His Asn Gly Ile Ala Asp Asn
930 935 940
Ala Thr Met Val Arg Val Asp Val Phe Glu Lys Gly Asp Lys Tyr Tyr
945 950 955 960
Leu Val Pro Ile Tyr Ser Trp Gln Val Ala Lys Gly Ile Leu Pro Asp
965 970 975
Arg Ala Val Val Gln Gly Lys Asp Glu Glu Asp Trp Gln Leu Ile Asp
980 985 990
Asp Ser Phe Asn Phe Lys Phe Ser Leu His Pro Asn Asp Leu Val Glu
995 1000 1005
Val Ile Thr Lys Lys Ala Arg Met Phe Gly Tyr Phe Ala Ser Cys
1010 1015 1020
His Arg Gly Thr Gly Asn Ile Asn Ile Arg Ile His Asp Leu Asp
1025 1030 1035
His Lys Ile Gly Lys Asn Gly Ile Leu Glu Gly Ile Gly Val Lys
1040 1045 1050
Thr Ala Leu Ser Phe Gln Lys Tyr Gln Ile Asp Glu Leu Gly Lys
1055 1060 1065
Glu Ile Arg Pro Cys Arg Leu Lys Lys Arg Pro Pro Val Arg
1070 1075 1080
<210> 31
<211> 1367
<212> PRT
<213> Artificial Sequence
<220>
<223> Cas9 from Streptococcus pyogenes
<400> 31
Met Asp Lys Lys Tyr Ser Ile Gly Leu Asp Ile Gly Thr Asn Ser Val
1 5 10 15
Gly Trp Ala Val Ile Thr Asp Asp Tyr Lys Val Pro Ser Lys Lys Phe
20 25 30
Lys Val Leu Gly Asn Thr Asp Arg His Ser Ile Lys Lys Asn Leu Ile
35 40 45
Gly Ala Leu Leu Phe Gly Ser Gly Glu Thr Ala Glu Ala Thr Arg Leu
50 55 60
Lys Arg Thr Ala Arg Arg Arg Tyr Thr Arg Arg Lys Asn Arg Ile Cys
65 70 75 80
Tyr Leu Gln Glu Ile Phe Ser Asn Glu Met Ala Lys Val Asp Asp Ser
85 90 95
Phe Phe His Arg Leu Glu Glu Ser Phe Leu Val Glu Glu Asp Lys Lys
100 105 110
His Glu Arg His Pro Ile Phe Gly Asn Ile Val Asp Glu Val Ala Tyr
115 120 125
His Glu Lys Tyr Pro Thr Ile Tyr His Leu Arg Lys Lys Leu Ala Asp
130 135 140
Ser Thr Asp Lys Ala Asp Leu Arg Leu Ile Tyr Leu Ala Leu Ala His
145 150 155 160
Met Ile Lys Phe Arg Gly His Phe Leu Ile Glu Gly Asp Leu Asn Pro
165 170 175
Asp Asn Ser Asp Val Asp Lys Leu Phe Ile Gln Leu Val Gln Ile Tyr
180 185 190
Asn Gln Leu Phe Glu Glu Asn Pro Ile Asn Ala Ser Arg Val Asp Ala
195 200 205
Lys Ala Ile Leu Ser Ala Arg Leu Ser Lys Ser Arg Arg Leu Glu Asn
210 215 220
Leu Ile Ala Gln Leu Pro Gly Glu Lys Arg Asn Gly Leu Phe Gly Asn
225 230 235 240
Leu Ile Ala Leu Ser Leu Gly Leu Thr Pro Asn Phe Lys Ser Asn Phe
245 250 255
Asp Leu Ala Glu Asp Ala Lys Leu Gln Leu Ser Lys Asp Thr Tyr Asp
260 265 270
Asp Asp Leu Asp Asn Leu Leu Ala Gln Ile Gly Asp Gln Tyr Ala Asp
275 280 285
Leu Phe Leu Ala Ala Lys Asn Leu Ser Asp Ala Ile Leu Leu Ser Asp
290 295 300
Ile Leu Arg Val Asn Ser Glu Ile Thr Lys Ala Pro Leu Ser Ala Ser
305 310 315 320
Met Ile Lys Arg Tyr Asp Glu His His Gln Asp Leu Thr Leu Leu Lys
325 330 335
Ala Leu Val Arg Gln Gln Leu Pro Glu Lys Tyr Lys Glu Ile Phe Phe
340 345 350
Asp Gln Ser Lys Asn Gly Tyr Ala Gly Tyr Ile Asp Gly Gly Ala Ser
355 360 365
Gln Glu Glu Phe Tyr Lys Phe Ile Lys Pro Ile Leu Glu Lys Met Asp
370 375 380
Gly Thr Glu Glu Leu Leu Val Lys Leu Asn Arg Glu Asp Leu Leu Arg
385 390 395 400
Lys Gln Arg Thr Phe Asp Asn Gly Ser Ile Pro His Gln Ile His Leu
405 410 415
Gly Glu Leu His Ala Ile Leu Arg Arg Gln Glu Asp Phe Tyr Pro Phe
420 425 430
Leu Lys Asp Asn Arg Glu Lys Ile Glu Lys Ile Leu Thr Phe Arg Ile
435 440 445
Pro Tyr Tyr Val Gly Pro Leu Ala Arg Gly Asn Ser Arg Phe Ala Trp
450 455 460
Met Thr Arg Lys Ser Glu Glu Thr Ile Thr Pro Trp Asn Phe Glu Glu
465 470 475 480
Val Val Asp Lys Gly Ala Ser Ala Gln Ser Phe Ile Glu Arg Met Thr
485 490 495
Asn Phe Asp Lys Asn Leu Pro Asn Glu Lys Val Leu Pro Lys His Ser
500 505 510
Leu Leu Tyr Glu Tyr Phe Thr Val Tyr Asn Glu Leu Thr Lys Val Lys
515 520 525
Tyr Val Thr Glu Gly Met Arg Lys Pro Ala Phe Leu Ser Gly Glu Gln
530 535 540
Lys Lys Ala Ile Val Asp Leu Leu Phe Lys Thr Asn Arg Lys Val Thr
545 550 555 560
Val Lys Gln Leu Lys Glu Asp Tyr Phe Lys Lys Ile Glu Cys Phe Asp
565 570 575
Ser Val Glu Ile Ser Gly Val Glu Asp Arg Phe Asn Ala Ser Leu Gly
580 585 590
Ala Tyr His Asp Leu Leu Lys Ile Ile Lys Asp Lys Asp Phe Leu Asp
595 600 605
Asn Glu Glu Asn Glu Asp Ile Leu Glu Asp Ile Val Leu Thr Leu Thr
610 615 620
Leu Phe Glu Asp Arg Gly Met Ile Glu Glu Arg Leu Lys Thr Tyr Ala
625 630 635 640
His Leu Phe Asp Asp Lys Val Met Lys Gln Leu Lys Arg Arg Arg Tyr
645 650 655
Thr Gly Trp Gly Arg Leu Ser Arg Lys Leu Ile Asn Gly Ile Arg Asp
660 665 670
Lys Gln Ser Gly Lys Thr Ile Leu Asp Phe Leu Lys Ser Asp Gly Phe
675 680 685
Ala Asn Arg Asn Phe Met Gln Leu Ile His Asp Asp Ser Leu Thr Phe
690 695 700
Lys Glu Asp Ile Gln Lys Ala Gln Val Ser Gly Gln Gly His Ser Leu
705 710 715 720
His Glu Gln Ile Ala Asn Leu Ala Gly Ser Pro Ala Ile Lys Lys Gly
725 730 735
Ile Leu Gln Thr Val Lys Ile Val Asp Glu Leu Val Lys Val Met Gly
740 745 750
His Lys Pro Glu Asn Ile Val Ile Glu Met Ala Arg Glu Asn Gln Thr
755 760 765
Thr Gln Lys Gly Gln Lys Asn Ser Arg Glu Arg Met Lys Arg Ile Glu
770 775 780
Glu Gly Ile Lys Glu Leu Gly Ser Gln Ile Leu Lys Glu His Pro Val
785 790 795 800
Glu Asn Thr Gln Leu Gln Asn Glu Lys Leu Tyr Leu Tyr Tyr Leu Gln
805 810 815
Asn Gly Arg Asp Met Tyr Val Asp Gln Glu Leu Asp Ile Asn Arg Leu
820 825 830
Ser Asp Tyr Asp Val Asp His Ile Val Pro Gln Ser Phe Ile Lys Asp
835 840 845
Asp Ser Ile Asp Asn Lys Val Leu Thr Arg Ser Asp Lys Asn Arg Gly
850 855 860
Lys Ser Asp Asn Val Pro Ser Glu Glu Val Val Lys Lys Met Lys Asn
865 870 875 880
Tyr Trp Arg Gln Leu Leu Asn Ala Lys Leu Ile Thr Gln Arg Lys Phe
885 890 895
Asp Asn Leu Thr Lys Ala Glu Arg Gly Gly Leu Ser Glu Leu Asp Lys
900 905 910
Ala Gly Phe Ile Lys Arg Gln Leu Val Glu Thr Arg Gln Ile Thr Lys
915 920 925
His Val Ala Gln Ile Leu Asp Ser Arg Met Asn Thr Lys Tyr Asp Glu
930 935 940
Asn Asp Lys Leu Ile Arg Glu Val Lys Val Ile Thr Leu Lys Ser Lys
945 950 955 960
Leu Val Ser Asp Phe Arg Lys Asp Phe Gln Phe Tyr Lys Val Arg Glu
965 970 975
Ile Asn Asn Tyr His His Ala His Asp Ala Tyr Leu Asn Ala Val Val
980 985 990
Gly Thr Ala Leu Ile Lys Lys Tyr Pro Lys Leu Glu Ser Glu Phe Val
995 1000 1005
Tyr Gly Asp Tyr Lys Val Tyr Asp Val Arg Lys Met Ile Ala Lys
1010 1015 1020
Ser Glu Gln Glu Ile Gly Lys Ala Thr Ala Lys Tyr Phe Phe Tyr
1025 1030 1035
Ser Asn Ile Met Asn Phe Phe Lys Thr Glu Ile Thr Leu Ala Asn
1040 1045 1050
Gly Glu Ile Arg Lys Arg Pro Leu Ile Glu Thr Asn Gly Glu Thr
1055 1060 1065
Gly Glu Ile Val Trp Asp Lys Gly Arg Asp Phe Ala Thr Val Arg
1070 1075 1080
Lys Val Leu Ser Met Pro Gln Val Asn Ile Val Lys Lys Thr Glu
1085 1090 1095
Val Gln Thr Gly Gly Phe Ser Lys Glu Ser Ile Leu Pro Lys Arg
1100 1105 1110
Asn Ser Asp Lys Leu Ile Ala Arg Lys Lys Asp Trp Asp Pro Lys
1115 1120 1125
Lys Tyr Gly Gly Phe Asp Ser Pro Thr Val Ala Tyr Ser Val Leu
1130 1135 1140
Val Val Ala Lys Val Glu Lys Gly Lys Ser Lys Lys Leu Lys Ser
1145 1150 1155
Val Lys Glu Leu Leu Gly Ile Thr Ile Met Glu Arg Ser Ser Phe
1160 1165 1170
Glu Lys Asn Pro Ile Asp Phe Leu Glu Ala Lys Gly Tyr Lys Glu
1175 1180 1185
Val Lys Lys Asp Leu Ile Ile Lys Leu Pro Lys Tyr Ser Leu Phe
1190 1195 1200
Glu Leu Glu Asn Gly Arg Lys Arg Met Leu Ala Ser Ala Gly Glu
1205 1210 1215
Leu Gln Lys Gly Asn Glu Leu Ala Leu Pro Ser Lys Tyr Val Asn
1220 1225 1230
Phe Leu Tyr Leu Ala Ser His Tyr Glu Lys Leu Lys Gly Ser Pro
1235 1240 1245
Glu Asp Asn Glu Gln Lys Gln Leu Phe Val Glu Gln His Lys His
1250 1255 1260
Tyr Leu Asp Glu Ile Ile Glu Gln Ile Ser Glu Phe Ser Lys Arg
1265 1270 1275
Val Ile Leu Ala Asp Ala Asn Leu Asp Lys Val Leu Ser Ala Tyr
1280 1285 1290
Asn Lys His Arg Asp Lys Pro Ile Arg Glu Gln Ala Glu Asn Ile
1295 1300 1305
Ile His Leu Phe Thr Leu Thr Asn Leu Gly Ala Pro Ala Ala Phe
1310 1315 1320
Lys Tyr Phe Asp Thr Thr Ile Asp Arg Lys Arg Tyr Thr Ser Thr
1325 1330 1335
Lys Glu Val Leu Asp Ala Thr Leu Ile His Gln Ser Ile Thr Gly
1340 1345 1350
Leu Tyr Glu Thr Arg Ile Asp Leu Ser Gln Leu Gly Gly Asp
1355 1360 1365
<210> 32
<211> 528
<212> PRT
<213> Artificial Sequence
<220>
<223> ZFP nucleic acid sequence
<400> 32
Ala Thr Gly Gly Cys Cys Cys Ala Gly Gly Cys Thr Gly Cys Thr Cys
1 5 10 15
Thr Thr Gly Ala Gly Cys Cys Cys Gly Gly Ala Gly Ala Gly Ala Ala
20 25 30
Ala Cys Cys Cys Thr Ala Cys Ala Ala Gly Thr Gly Cys Cys Cys Gly
35 40 45
Gly Ala Gly Thr Gly Cys Gly Gly Ala Ala Ala Gly Thr Cys Cys Thr
50 55 60
Thr Cys Thr Cys Thr Gly Ala Gly Cys Gly Gly Ala Gly Thr Cys Ala
65 70 75 80
Cys Cys Thr Cys Cys Gly Ala Gly Ala Gly Cys Ala Cys Cys Ala Gly
85 90 95
Cys Gly Gly Ala Cys Thr Cys Ala Thr Ala Cys Gly Gly Gly Cys Gly
100 105 110
Ala Ala Ala Ala Ala Cys Cys Ala Thr Ala Cys Ala Ala Gly Thr Gly
115 120 125
Cys Cys Cys Ala Gly Ala Ala Thr Gly Thr Gly Gly Thr Ala Ala Ala
130 135 140
Thr Cys Thr Thr Thr Thr Thr Cys Thr Cys Gly Gly Gly Cys Thr Gly
145 150 155 160
Ala Cys Ala Ala Cys Cys Thr Gly Ala Cys Thr Gly Ala Ala Cys Ala
165 170 175
Thr Cys Ala Gly Cys Gly Cys Ala Cys Gly Cys Ala Cys Ala Cys Cys
180 185 190
Gly Gly Thr Gly Ala Ala Ala Ala Ala Cys Cys Thr Thr Ala Cys Ala
195 200 205
Ala Gly Thr Gly Thr Cys Cys Ala Gly Ala Gly Thr Gly Thr Gly Gly
210 215 220
Cys Ala Ala Gly Ala Gly Cys Thr Thr Thr Thr Cys Thr Ala Gly Thr
225 230 235 240
Ala Gly Ala Ala Gly Gly Ala Cys Cys Thr Gly Thr Cys Gly Ala Gly
245 250 255
Cys Gly Cys Ala Thr Cys Ala Gly Cys Gly Gly Ala Cys Thr Cys Ala
260 265 270
Cys Ala Cys Cys Gly Gly Cys Gly Ala Ala Ala Ala Ala Cys Cys Cys
275 280 285
Thr Ala Thr Ala Ala Gly Thr Gly Thr Cys Cys Gly Gly Ala Ala Thr
290 295 300
Gly Thr Gly Gly Ala Ala Ala Gly Ala Gly Cys Thr Thr Thr Ala Gly
305 310 315 320
Cys Cys Gly Cys Ala Ala Cys Gly Ala Cys Ala Cys Cys Cys Thr Thr
325 330 335
Ala Cys Thr Gly Ala Ala Cys Ala Cys Cys Ala Gly Cys Gly Ala Ala
340 345 350
Cys Ala Cys Ala Cys Ala Cys Gly Gly Gly Ala Gly Ala Ala Ala Ala
355 360 365
Ala Cys Cys Ala Thr Ala Thr Ala Ala Ala Thr Gly Thr Cys Cys Gly
370 375 380
Gly Ala Ala Thr Gly Thr Gly Gly Cys Ala Ala Ala Ala Gly Thr Thr
385 390 395 400
Thr Thr Ala Gly Thr Cys Gly Gly Ala Gly Thr Gly Ala Thr Ala Ala
405 410 415
Ala Cys Thr Thr Ala Cys Gly Gly Ala Gly Cys Ala Cys Cys Ala Ala
420 425 430
Cys Gly Gly Ala Cys Ala Cys Ala Cys Ala Cys Cys Gly Gly Ala Gly
435 440 445
Ala Gly Ala Ala Gly Cys Cys Ala Thr Ala Thr Ala Ala Gly Thr Gly
450 455 460
Thr Cys Cys Thr Gly Ala Ala Thr Gly Thr Gly Gly Ala Ala Ala Gly
465 470 475 480
Thr Cys Cys Thr Thr Cys Thr Cys Ala Cys Ala Gly Cys Thr Thr Gly
485 490 495
Cys Thr Cys Ala Thr Cys Thr Gly Cys Gly Ala Gly Cys Ala Cys Ala
500 505 510
Thr Cys Ala Gly Cys Gly Cys Ala Cys Ala Cys Ala Cys Ala Cys Cys
515 520 525
<210> 33
<211> 176
<212> PRT
<213> Artificial Sequence
<220>
<223> ZFP amino acid sequence
<400> 33
Met Ala Gln Ala Ala Leu Glu Pro Gly Glu Lys Pro Tyr Lys Cys Pro
1 5 10 15
Glu Cys Gly Lys Ser Phe Ser Glu Arg Ser His Leu Arg Glu His Gln
20 25 30
Arg Thr His Thr Gly Glu Lys Pro Tyr Lys Cys Pro Glu Cys Gly Lys
35 40 45
Ser Phe Ser Arg Ala Asp Asn Leu Thr Glu His Gln Arg Thr His Thr
50 55 60
Gly Glu Lys Pro Tyr Lys Cys Pro Glu Cys Gly Lys Ser Phe Ser Ser
65 70 75 80
Arg Arg Thr Cys Arg Ala His Gln Arg Thr His Thr Gly Glu Lys Pro
85 90 95
Tyr Lys Cys Pro Glu Cys Gly Lys Ser Phe Ser Arg Asn Asp Thr Leu
100 105 110
Thr Glu His Gln Arg Thr His Thr Gly Glu Lys Pro Tyr Lys Cys Pro
115 120 125
Glu Cys Gly Lys Ser Phe Ser Arg Ser Asp Lys Leu Thr Glu His Gln
130 135 140
Arg Thr His Thr Gly Glu Lys Pro Tyr Lys Cys Pro Glu Cys Gly Lys
145 150 155 160
Ser Phe Ser Gln Leu Ala His Leu Arg Ala His Gln Arg Thr His Thr
165 170 175
<210> 34
<211> 555
<212> DNA
<213> Artificial Sequence
<220>
<223> ZNF-E2C Nucleotide
<400> 34
atggcgcagg cggcgctgga accgggcgaa aaaccgtata aatgcccgga atgcggcaaa 60
agctttagcc gcaaagatag cctggtgcgc catcagcgca cccataccgg cgaaaaaccg 120
tataaatgcc cggaatgcgg caaaagcttt agccagagcg gcgatctgcg ccgccatcag 180
cgcacccata ccggcgaaaa accgtataaa tgcccggaat gcggcaaaag ctttagcgat 240
tgccgcgatc tggcgcgcca tcagcgcacc cataccggcg aaaaaccgta taaatgcccg 300
gaatgcggca aaagctttag ccagagcagc catctggtgc gccatcagcg cacccatacc 360
ggcgaaaaac cgtataaatg cccggaatgc ggcaaaagct ttagcgattg ccgcgatctg 420
gcgcgccatc agcgcaccca taccggcgaa aaaccgtata aatgcccgga atgcggcaaa 480
agctttagcc gcagcgataa actggtgcgc catcagcgca cccataccgg caaaaaaacc 540
agcggccagg cgggc 555
<210> 35
<211> 185
<212> PRT
<213> Artificial Sequence
<220>
<223> ZNF-E2C Aminoacid
<400> 35
Met Ala Gln Ala Ala Leu Glu Pro Gly Glu Lys Pro Tyr Lys Cys Pro
1 5 10 15
Glu Cys Gly Lys Ser Phe Ser Arg Lys Asp Ser Leu Val Arg His Gln
20 25 30
Arg Thr His Thr Gly Glu Lys Pro Tyr Lys Cys Pro Glu Cys Gly Lys
35 40 45
Ser Phe Ser Gln Ser Gly Asp Leu Arg Arg His Gln Arg Thr His Thr
50 55 60
Gly Glu Lys Pro Tyr Lys Cys Pro Glu Cys Gly Lys Ser Phe Ser Asp
65 70 75 80
Cys Arg Asp Leu Ala Arg His Gln Arg Thr His Thr Gly Glu Lys Pro
85 90 95
Tyr Lys Cys Pro Glu Cys Gly Lys Ser Phe Ser Gln Ser Ser His Leu
100 105 110
Val Arg His Gln Arg Thr His Thr Gly Glu Lys Pro Tyr Lys Cys Pro
115 120 125
Glu Cys Gly Lys Ser Phe Ser Asp Cys Arg Asp Leu Ala Arg His Gln
130 135 140
Arg Thr His Thr Gly Glu Lys Pro Tyr Lys Cys Pro Glu Cys Gly Lys
145 150 155 160
Ser Phe Ser Arg Ser Asp Lys Leu Val Arg His Gln Arg Thr His Thr
165 170 175
Gly Lys Lys Thr Ser Gly Gln Ala Gly
180 185
<210> 36
<211> 555
<212> DNA
<213> Artificial Sequence
<220>
<223> ZNF-E3 Nucleotide
<400> 36
atggcgcagg cggcgctgga accgggcgaa aaaccgtata aatgcccgga atgcggcaaa 60
agctttagcg atccgggcgc gctggtgcgc catcagcgca cccataccgg cgaaaaaccg 120
tataaatgcc cggaatgcgg caaaagcttt agccagagca gccatctggt gcgccatcag 180
cgcacccata ccggcgaaaa accgtataaa tgcccggaat gcggcaaaag ctttagcgat 240
tgccgcgatc tggcgcgcca tcagcgcacc cataccggcg aaaaaccgta taaatgcccg 300
gaatgcggca aaagctttag ccagagcagc catctggtgc gccatcagcg cacccatacc 360
ggcgaaaaac cgtataaatg cccggaatgc ggcaaaagct ttagcgattg ccgcgatctg 420
gcgcgccatc agcgcaccca taccggcgaa aaaccgtata aatgcccgga atgcggcaaa 480
agctttagcc agagcagcca tctggtgcgc catcagcgca cccataccgg caaaaaaacc 540
agcggccagg cgggc 555
<210> 37
<211> 185
<212> PRT
<213> Artificial Sequence
<220>
<223> ZNF-E3 Amino acid
<400> 37
Met Ala Gln Ala Ala Leu Glu Pro Gly Glu Lys Pro Tyr Lys Cys Pro
1 5 10 15
Glu Cys Gly Lys Ser Phe Ser Asp Pro Gly Ala Leu Val Arg His Gln
20 25 30
Arg Thr His Thr Gly Glu Lys Pro Tyr Lys Cys Pro Glu Cys Gly Lys
35 40 45
Ser Phe Ser Gln Ser Ser His Leu Val Arg His Gln Arg Thr His Thr
50 55 60
Gly Glu Lys Pro Tyr Lys Cys Pro Glu Cys Gly Lys Ser Phe Ser Asp
65 70 75 80
Cys Arg Asp Leu Ala Arg His Gln Arg Thr His Thr Gly Glu Lys Pro
85 90 95
Tyr Lys Cys Pro Glu Cys Gly Lys Ser Phe Ser Gln Ser Ser His Leu
100 105 110
Val Arg His Gln Arg Thr His Thr Gly Glu Lys Pro Tyr Lys Cys Pro
115 120 125
Glu Cys Gly Lys Ser Phe Ser Asp Cys Arg Asp Leu Ala Arg His Gln
130 135 140
Arg Thr His Thr Gly Glu Lys Pro Tyr Lys Cys Pro Glu Cys Gly Lys
145 150 155 160
Ser Phe Ser Gln Ser Ser His Leu Val Arg His Gln Arg Thr His Thr
165 170 175
Gly Lys Lys Thr Ser Gly Gln Ala Gly
180 185
<210> 38
<211> 528
<212> PRT
<213> Artificial Sequence
<220>
<223> ZNF-TRCa Nucleotide
<400> 38
Ala Thr Gly Gly Cys Gly Cys Ala Gly Gly Cys Gly Gly Cys Thr Cys
1 5 10 15
Thr Thr Gly Ala Ala Cys Cys Cys Gly Gly Gly Gly Ala Gly Ala Ala
20 25 30
Ala Cys Cys Cys Thr Ala Thr Ala Ala Ala Thr Gly Cys Cys Cys Thr
35 40 45
Gly Ala Gly Thr Gly Thr Gly Gly Cys Ala Ala Gly Ala Gly Thr Thr
50 55 60
Thr Thr Thr Cys Ala Ala Cys Cys Ala Cys Ala Gly Gly Ala Ala Ala
65 70 75 80
Cys Thr Thr Gly Ala Cys Ala Gly Thr Cys Cys Ala Cys Cys Ala Ala
85 90 95
Cys Gly Gly Ala Cys Cys Cys Ala Cys Ala Cys Cys Gly Gly Cys Gly
100 105 110
Ala Gly Ala Ala Ala Cys Cys Ala Thr Ala Cys Ala Ala Gly Thr Gly
115 120 125
Thr Cys Cys Gly Gly Ala Gly Thr Gly Thr Gly Gly Thr Ala Ala Gly
130 135 140
Thr Cys Thr Thr Thr Cys Thr Cys Ala Ala Gly Thr Cys Cys Thr Gly
145 150 155 160
Cys Cys Gly Ala Cys Cys Thr Thr Ala Cys Cys Ala Gly Ala Cys Ala
165 170 175
Thr Cys Ala Ala Cys Gly Cys Ala Cys Ala Cys Ala Thr Ala Cys Ala
180 185 190
Gly Gly Thr Gly Ala Ala Ala Ala Ala Cys Cys Thr Thr Ala Cys Ala
195 200 205
Ala Gly Thr Gly Cys Cys Cys Ala Gly Ala Gly Thr Gly Cys Gly Gly
210 215 220
Ala Ala Ala Ala Ala Gly Thr Thr Thr Thr Thr Cys Ala Cys Ala Ala
225 230 235 240
Thr Cys Thr Gly Gly Cys Gly Ala Cys Cys Thr Cys Cys Gly Cys Ala
245 250 255
Gly Gly Cys Ala Cys Cys Ala Gly Cys Gly Cys Ala Cys Thr Cys Ala
260 265 270
Cys Ala Cys Cys Gly Gly Thr Gly Ala Ala Ala Ala Ala Cys Cys Ala
275 280 285
Thr Ala Cys Ala Ala Gly Thr Gly Thr Cys Cys Thr Gly Ala Gly Thr
290 295 300
Gly Cys Gly Gly Gly Ala Ala Gly Ala Gly Thr Thr Thr Thr Ala Gly
305 310 315 320
Thr Cys Ala Ala Cys Gly Ala Gly Cys Thr Cys Ala Thr Cys Thr Gly
325 330 335
Gly Ala Gly Cys Gly Ala Cys Ala Cys Cys Ala Ala Ala Gly Gly Ala
340 345 350
Cys Thr Cys Ala Thr Ala Cys Thr Gly Gly Gly Gly Ala Gly Ala Ala
355 360 365
Ala Cys Cys Gly Thr Ala Cys Ala Ala Ala Thr Gly Thr Cys Cys Cys
370 375 380
Gly Ala Ala Thr Gly Thr Gly Gly Gly Ala Ala Gly Ala Gly Cys Thr
385 390 395 400
Thr Cys Thr Cys Thr Ala Cys Cys Ala Ala Gly Ala Ala Thr Thr Cys
405 410 415
Cys Cys Thr Thr Ala Cys Ala Gly Ala Gly Cys Ala Cys Cys Ala Gly
420 425 430
Cys Gly Cys Ala Cys Gly Cys Ala Thr Ala Cys Gly Gly Gly Ala Gly
435 440 445
Ala Gly Ala Ala Gly Cys Cys Gly Thr Ala Thr Ala Ala Gly Thr Gly
450 455 460
Thr Cys Cys Gly Gly Ala Ala Thr Gly Thr Gly Gly Cys Ala Ala Gly
465 470 475 480
Ala Gly Cys Thr Thr Thr Thr Cys Cys Ala Gly Ala Ala Gly Thr Gly
485 490 495
Ala Cys Cys Ala Cys Cys Thr Thr Ala Cys Ala Ala Cys Cys Cys Ala
500 505 510
Cys Cys Ala Gly Ala Gly Gly Ala Cys Gly Cys Ala Cys Ala Cys Cys
515 520 525
<210> 39
<211> 176
<212> PRT
<213> Artificial Sequence
<220>
<223> ZNF-TRCa Aminoacid
<400> 39
Met Ala Gln Ala Ala Leu Glu Pro Gly Glu Lys Pro Tyr Lys Cys Pro
1 5 10 15
Glu Cys Gly Lys Ser Phe Ser Thr Thr Gly Asn Leu Thr Val His Gln
20 25 30
Arg Thr His Thr Gly Glu Lys Pro Tyr Lys Cys Pro Glu Cys Gly Lys
35 40 45
Ser Phe Ser Ser Pro Ala Asp Leu Thr Arg His Gln Arg Thr His Thr
50 55 60
Gly Glu Lys Pro Tyr Lys Cys Pro Glu Cys Gly Lys Ser Phe Ser Gln
65 70 75 80
Ser Gly Asp Leu Arg Arg His Gln Arg Thr His Thr Gly Glu Lys Pro
85 90 95
Tyr Lys Cys Pro Glu Cys Gly Lys Ser Phe Ser Gln Arg Ala His Leu
100 105 110
Glu Arg His Gln Arg Thr His Thr Gly Glu Lys Pro Tyr Lys Cys Pro
115 120 125
Glu Cys Gly Lys Ser Phe Ser Thr Lys Asn Ser Leu Thr Glu His Gln
130 135 140
Arg Thr His Thr Gly Glu Lys Pro Tyr Lys Cys Pro Glu Cys Gly Lys
145 150 155 160
Ser Phe Ser Arg Ser Asp His Leu Thr Thr His Gln Arg Thr His Thr
165 170 175
<210> 40
<211> 18
<212> PRT
<213> Artificial Sequence
<220>
<223> AAVS1 site
<400> 40
Ala Gly Ala Cys Gly Gly Cys Cys Gly Cys Gly Thr Cys Ala Gly Ala
1 5 10 15
Gly Cys
<210> 41
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Zinc Finger 1
<400> 41
Glu Arg Ser His Leu Arg Glu
1 5
<210> 42
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Zinc Finger 2
<400> 42
Arg Ala Asp Asn Leu Thr Glu
1 5
<210> 43
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Zinc Finger 3
<400> 43
Ser Arg Arg Thr Cys Arg Ala
1 5
<210> 44
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Zinc Finger 4
<400> 44
Arg Asn Asp Thr Leu Thr Glu
1 5
<210> 45
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Zinc Finger 5
<400> 45
Arg Ser Asp Lys Leu Thr Glu
1 5
<210> 46
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Zinc Finger 6
<400> 46
Gln Leu Ala His Leu Arg Ala
1 5
<210> 47
<211> 51
<212> DNA
<213> Artificial Sequence
<220>
<223> Nuclear Localization Signal
<400> 47
atggctccaa agaaaaagag gaaagtggga atccacggag tccccgccgc t 51
<210> 48
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> GGS linker Nucleic Acid
<400> 48
ggtggatctg gcggtggatc tggtggcggt 30
<210> 49
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> GGS linker Amino Acid
<400> 49
Gly Gly Ser Gly Gly Gly Ser Gly Gly Gly
1 5 10
<210> 50
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> GGS4x Linker Nucleic Acid
<400> 50
ggagggagtg gtgggtccgg tggtagtggc ggatcc 36
<210> 51
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> GGS4x Linker Amino Acid
<400> 51
Gly Gly Ser Gly Gly Ser Gly Gly Ser Gly Gly Ser
1 5 10
<210> 52
<211> 45
<212> DNA
<213> Artificial Sequence
<220>
<223> GGS5x Linker Nucleic Acid
<400> 52
ggaggctccg gtgggtctgg tgggagcggt ggtagtggcg gatcc 45
<210> 53
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> GGS5x Linker Amino Acid
<400> 53
Gly Gly Ser Gly Gly Ser Gly Gly Ser Gly Gly Ser Gly Gly Ser
1 5 10 15
<210> 54
<211> 54
<212> DNA
<213> Artificial Sequence
<220>
<223> GGS6x Linker Nucleic Acid
<400> 54
ggaggcagtg gtgggagcgg tggttccggg ggtagtggtg gttccggggg atcc 54
<210> 55
<211> 18
<212> PRT
<213> Artificial Sequence
<220>
<223> GGS6x Linker Amino Acid
<400> 55
Gly Gly Ser Gly Gly Ser Gly Gly Ser Gly Gly Ser Gly Gly Ser Gly
1 5 10 15
Gly Ser
<210> 56
<211> 63
<212> DNA
<213> Artificial Sequence
<220>
<223> GGS7x Linker Nucleic Acid
<400> 56
ggaggttctg gaggctccgg tgggtccggg ggaagtgggg ggtcaggcgg atcaggagga 60
tcc 63
<210> 57
<211> 21
<212> PRT
<213> Artificial Sequence
<220>
<223> GGS7x Linker Amino Acid
<400> 57
Gly Gly Ser Gly Gly Ser Gly Gly Ser Gly Gly Ser Gly Gly Ser Gly
1 5 10 15
Gly Ser Gly Gly Ser
20
<210> 58
<211> 75
<212> DNA
<213> Artificial Sequence
<220>
<223> GGS8x Linker Nucleic Acid
<400> 58
ggaggtagcg gaggttccgg agggagcggc gggagtgggg gaagcggggg aagtggagga 60
tccgggggag gatcc 75
<210> 59
<211> 21
<212> PRT
<213> Artificial Sequence
<220>
<223> GGS8x Linker Amino Acid
<400> 59
Gly Gly Ser Gly Gly Ser Gly Gly Ser Gly Gly Ser Gly Gly Ser Gly
1 5 10 15
Gly Ser Gly Gly Ser
20
<210> 60
<211> 48
<212> DNA
<213> Artificial Sequence
<220>
<223> Linker XTEN Nucleic Acid
<400> 60
tccggtagcg aaacaccggg gacttcagaa tcggccaccc cggagtct 48
<210> 61
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> Linker XTEN Amino Acid
<400> 61
Ser Gly Ser Glu Thr Pro Gly Thr Ser Glu Ser Ala Thr Pro Glu Ser
1 5 10 15
<210> 62
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Linker B Nucleic Acid
<400> 62
ggaagcgccg gtagtgcggc tgggtctggc gagttc 36
<210> 63
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Linker B Amino Acid
<400> 63
Gly Ser Ala Gly Ser Ala Ala Gly Ser Gly Glu Phe
1 5 10
<210> 64
<211> 4104
<212> DNA
<213> Artificial Sequence
<220>
<223> human Cas9 (hCas9) nucleic acid
<400> 64
atggacaaga agtactccat tgggctcgat atcggcacaa acagcgtcgg ctgggccgtc 60
attacggacg agtacaaggt gccgagcaaa aaattcaaag ttctgggcaa taccgatcgc 120
cacagcataa agaagaacct cattggcgcc ctcctgttcg actccgggga gacggccgaa 180
gccacgcggc tcaaaagaac agcacggcgc agatataccc gcagaaagaa tcggatctgc 240
tacctgcagg agatctttag taatgagatg gctaaggtgg atgactcttt cttccatagg 300
ctggaggagt cctttttggt ggaggaggat aaaaagcacg agcgccaccc aatctttggc 360
aatatcgtgg acgaggtggc gtaccatgaa aagtacccaa ccatatatca tctgaggaag 420
aagcttgtag acagtactga taaggctgac ttgcggttga tctatctcgc gctggcgcat 480
atgatcaaat ttcggggaca cttcctcatc gagggggacc tgaacccaga caacagcgat 540
gtcgacaaac tctttatcca actggttcag acttacaatc agcttttcga agagaacccg 600
atcaacgcat ccggagttga cgccaaagca atcctgagcg ctaggctgtc caaatcccgg 660
cggctcgaaa acctcatcgc acagctccct ggggagaaga agaacggcct gtttggtaat 720
cttatcgccc tgtcactcgg gctgaccccc aactttaaat ctaacttcga cctggccgaa 780
gatgccaagc ttcaactgag caaagacacc tacgatgatg atctcgacaa tctgctggcc 840
cagatcggcg accagtacgc agaccttttt ttggcggcaa agaacctgtc agacgccatt 900
ctgctgagtg atattctgcg agtgaacacg gagatcacca aagctccgct gagcgctagt 960
atgatcaagc gctatgatga gcaccaccaa gacttgactt tgctgaaggc ccttgtcaga 1020
cagcaactgc ctgagaagta caaggaaatt ttcttcgatc agtctaaaaa tggctacgcc 1080
ggatacattg acggcggagc aagccaggag gaattttaca aatttattaa gcccatcttg 1140
gaaaaaatgg acggcaccga ggagctgctg gtaaagctta acagagaaga tctgttgcgc 1200
aaacagcgca ctttcgacaa tggaagcatc ccccaccaga ttcacctggg cgaactgcac 1260
gctatcctca ggcggcaaga ggatttctac ccctttttga aagataacag ggaaaagatt 1320
gagaaaatcc tcacatttcg gataccctac tatgtaggcc ccctcgcccg gggaaattcc 1380
agattcgcgt ggatgactcg caaatcagaa gagaccatca ctccctggaa cttcgaggaa 1440
gtcgtggata agggggcctc tgcccagtcc ttcatcgaaa ggatgactaa ctttgataaa 1500
aatctgccta acgaaaaggt gcttcctaaa cactctctgc tgtacgagta cttcacagtt 1560
tataacgagc tcaccaaggt caaatacgtc acagaaggga tgagaaagcc agcattcctg 1620
tctggagagc agaagaaagc tatcgtggac ctcctcttca agacgaaccg gaaagttacc 1680
gtgaaacagc tcaaagaaga ctatttcaaa aagattgaat gtttcgactc tgttgaaatc 1740
agcggagtgg aggatcgctt caacgcatcc ctgggaacgt atcacgatct cctgaaaatc 1800
attaaagaca aggacttcct ggacaatgag gagaacgagg acattcttga ggacattgtc 1860
ctcaccctta cgttgtttga agatagggag atgattgaag aacgcttgaa aacttacgct 1920
catctcttcg acgacaaagt catgaaacag ctcaagaggc gccgatatac aggatggggg 1980
cggctgtcaa gaaaactgat caatgggatc cgagacaagc agagtggaaa gacaatcctg 2040
gattttctta agtccgatgg atttgccaac cggaacttca tgcagttgat ccatgatgac 2100
tctctcacct ttaaggagga catccagaaa gcacaagttt ctggccaggg ggacagtctt 2160
cacgagcaca tcgctaatct tgcaggtagc ccagctatca aaaagggaat actgcagacc 2220
gttaaggtcg tggatgaact cgtcaaagta atgggaaggc ataagcccga gaatatcgtt 2280
atcgagatgg cccgagagaa ccaaactacc cagaagggac agaagaacag tagggaaagg 2340
atgaagagga ttgaagaggg tataaaagaa ctggggtccc aaatccttaa ggaacaccca 2400
gttgaaaaca cccagcttca gaatgagaag ctctacctgt actacctgca gaacggcagg 2460
gacatgtacg tggatcagga actggacatc aatcggctct ccgactacga cgtggatcat 2520
atcgtgcccc agtcttttct caaagatgat tctattgata ataaagtgtt gacaagatcc 2580
gataaaaata gagggaagag tgataacgtc ccctcagaag aagttgtcaa gaaaatgaaa 2640
aattattggc ggcagctgct gaacgccaaa ctgatcacac aacggaagtt cgataatctg 2700
actaaggctg aacgaggtgg cctgtctgag ttggataaag ccggcttcat caaaaggcag 2760
cttgttgaga cacgccagat caccaagcac gtggcccaaa ttctcgattc acgcatgaac 2820
accaagtacg atgaaaatga caaactgatt cgagaggtga aagttattac tctgaagtct 2880
aagctggtct cagatttcag aaaggacttt cagttttata aggtgagaga gatcaacaat 2940
taccaccatg cgcatgatgc ctacctgaat gcagtggtag gcactgcact tatcaaaaaa 3000
tatcccaagc ttgaatctga atttgtttac ggagactata aagtgtacga tgttaggaaa 3060
atgatcgcaa agtctgagca ggaaataggc aaggccaccg ctaagtactt cttttacagc 3120
aatattatga attttttcaa gaccgagatt acactggcca atggagagat tcggaagcga 3180
ccacttatcg aaacaaacgg agaaacagga gaaatcgtgt gggacaaggg tagggatttc 3240
gcgacagtcc ggaaggtcct gtccatgccg caggtgaaca tcgttaaaaa gaccgaagta 3300
cagaccggag gcttctccaa ggaaagtatc ctcccgaaaa ggaacagcga caagctgatc 3360
gcacgcaaaa aagattggga ccccaagaaa tacggcggat tcgattctcc tacagtcgct 3420
tacagtgtac tggttgtggc caaagtggag aaagggaagt ctaaaaaact caaaagcgtc 3480
aaggaactgc tgggcatcac aatcatggag cgatcaagct tcgaaaaaaa ccccatcgac 3540
tttctcgagg cgaaaggata taaagaggtc aaaaaagacc tcatcattaa gcttcccaag 3600
tactctctct ttgagcttga aaacggccgg aaacgaatgc tcgctagtgc gggcgagctg 3660
cagaaaggta acgagctggc actgccctct aaatacgtta atttcttgta tctggccagc 3720
cactatgaaa agctcaaagg gtctcccgaa gataatgagc agaagcagct gttcgtggaa 3780
caacacaaac actaccttga tgagatcatc gagcaaataa gcgaattctc caaaagagtg 3840
atcctcgccg acgctaacct cgataaggtg ctttctgctt acaataagca cagggataag 3900
cccatcaggg agcaggcaga aaacattatc cacttgttta ctctgaccaa cttgggcgcg 3960
cctgcagcct tcaagtactt cgacaccacc atagacagaa agcggtacac ctctacaaag 4020
gaggtcctgg acgccacact gattcatcag tcaattacgg ggctctatga aacaagaatc 4080
gacctctctc agctcggtgg agac 4104
<210> 65
<211> 4104
<212> DNA
<213> Artificial Sequence
<220>
<223> nickase Cas9 (nCas9) nucleic acid
<400> 65
atggacaaga agtactccat tgggctcgct atcggcacaa acagcgtcgg ctgggccgtc 60
attacggacg agtacaaggt gccgagcaaa aaattcaaag ttctgggcaa taccgatcgc 120
cacagcataa agaagaacct cattggcgcc ctcctgttcg actccgggga gacggccgaa 180
gccacgcggc tcaaaagaac agcacggcgc agatataccc gcagaaagaa tcggatctgc 240
tacctgcagg agatctttag taatgagatg gctaaggtgg atgactcttt cttccatagg 300
ctggaggagt cctttttggt ggaggaggat aaaaagcacg agcgccaccc aatctttggc 360
aatatcgtgg acgaggtggc gtaccatgaa aagtacccaa ccatatatca tctgaggaag 420
aagcttgtag acagtactga taaggctgac ttgcggttga tctatctcgc gctggcgcat 480
atgatcaaat ttcggggaca cttcctcatc gagggggacc tgaacccaga caacagcgat 540
gtcgacaaac tctttatcca actggttcag acttacaatc agcttttcga agagaacccg 600
atcaacgcat ccggagttga cgccaaagca atcctgagcg ctaggctgtc caaatcccgg 660
cggctcgaaa acctcatcgc acagctccct ggggagaaga agaacggcct gtttggtaat 720
cttatcgccc tgtcactcgg gctgaccccc aactttaaat ctaacttcga cctggccgaa 780
gatgccaagc ttcaactgag caaagacacc tacgatgatg atctcgacaa tctgctggcc 840
cagatcggcg accagtacgc agaccttttt ttggcggcaa agaacctgtc agacgccatt 900
ctgctgagtg atattctgcg agtgaacacg gagatcacca aagctccgct gagcgctagt 960
atgatcaagc gctatgatga gcaccaccaa gacttgactt tgctgaaggc ccttgtcaga 1020
cagcaactgc ctgagaagta caaggaaatt ttcttcgatc agtctaaaaa tggctacgcc 1080
ggatacattg acggcggagc aagccaggag gaattttaca aatttattaa gcccatcttg 1140
gaaaaaatgg acggcaccga ggagctgctg gtaaagctta acagagaaga tctgttgcgc 1200
aaacagcgca ctttcgacaa tggaagcatc ccccaccaga ttcacctggg cgaactgcac 1260
gctatcctca ggcggcaaga ggatttctac ccctttttga aagataacag ggaaaagatt 1320
gagaaaatcc tcacatttcg gataccctac tatgtaggcc ccctcgcccg gggaaattcc 1380
agattcgcgt ggatgactcg caaatcagaa gagaccatca ctccctggaa cttcgaggaa 1440
gtcgtggata agggggcctc tgcccagtcc ttcatcgaaa ggatgactaa ctttgataaa 1500
aatctgccta acgaaaaggt gcttcctaaa cactctctgc tgtacgagta cttcacagtt 1560
tataacgagc tcaccaaggt caaatacgtc acagaaggga tgagaaagcc agcattcctg 1620
tctggagagc agaagaaagc tatcgtggac ctcctcttca agacgaaccg gaaagttacc 1680
gtgaaacagc tcaaagaaga ctatttcaaa aagattgaat gtttcgactc tgttgaaatc 1740
agcggagtgg aggatcgctt caacgcatcc ctgggaacgt atcacgatct cctgaaaatc 1800
attaaagaca aggacttcct ggacaatgag gagaacgagg acattcttga ggacattgtc 1860
ctcaccctta cgttgtttga agatagggag atgattgaag aacgcttgaa aacttacgct 1920
catctcttcg acgacaaagt catgaaacag ctcaagaggc gccgatatac aggatggggg 1980
cggctgtcaa gaaaactgat caatgggatc cgagacaagc agagtggaaa gacaatcctg 2040
gattttctta agtccgatgg atttgccaac aggaacttca tgcagttgat ccatgatgac 2100
tctctcacct ttaaggagga catccagaaa gcacaagttt ctggccaggg ggacagtctt 2160
cacgagcaca tcgctaatct tgcaggtagc ccagctatca aaaagggaat actgcagacc 2220
gttaaggtcg tggatgaact cgtcaaagta atgggaaggc ataagcccga gaatatcgtt 2280
atcgagatgg cccgagagaa ccaaactacc cagaagggac agaagaacag tagggaaagg 2340
atgaagagga ttgaagaggg tataaaagaa ctggggtccc aaatccttaa ggaacaccca 2400
gttgaaaaca cccagcttca gaatgagaag ctctacctgt actacctgca gaacggcagg 2460
gacatgtacg tggatcagga actggacatc aatcggctct ccgactacga cgtggatcat 2520
atcgtgcccc agtcttttct caaagatgat tctattgata ataaagtgtt gacaagatcc 2580
gataaaaata gagggaagag tgataacgtc ccctcagaag aagttgtcaa gaaaatgaaa 2640
aattattggc ggcagctgct gaacgccaaa ctgatcacac aacggaagtt cgataatctg 2700
actaaggctg aacgaggtgg cctgtctgag ttggataaag ccggcttcat caaaaggcag 2760
cttgttgaga cacgccagat caccaagcac gtggcccaaa ttctcgattc acgcatgaac 2820
accaagtacg atgaaaatga caaactgatt cgagaggtga aagttattac tctgaagtct 2880
aagctggtct cagatttcag aaaggacttt cagttttata aggtgagaga gatcaacaat 2940
taccaccatg cgcatgatgc ctacctgaat gcagtggtag gcactgcact tatcaaaaaa 3000
tatcccaagc ttgaatctga atttgtttac ggagactata aagtgtacga tgttaggaaa 3060
atgatcgcaa agtctgagca ggaaataggc aaggccaccg ctaagtactt cttttacagc 3120
aatattatga attttttcaa gaccgagatt acactggcca atggagagat tcggaagcga 3180
ccacttatcg aaacaaacgg agaaacagga gaaatcgtgt gggacaaggg tagggatttc 3240
gcgacagtcc ggaaggtcct gtccatgccg caggtgaaca tcgttaaaaa gaccgaagta 3300
cagaccggag gcttctccaa ggaaagtatc ctcccgaaaa ggaacagcga caagctgatc 3360
gcacgcaaaa aagattggga ccccaagaaa tacggcggat tcgattctcc tacagtcgct 3420
tacagtgtac tggttgtggc caaagtggag aaagggaagt ctaaaaaact caaaagcgtc 3480
aaggaactgc tgggcatcac aatcatggag cgatcaagct tcgaaaaaaa ccccatcgac 3540
tttctcgagg cgaaaggata taaagaggtc aaaaaagacc tcatcattaa gcttcccaag 3600
tactctctct ttgagcttga aaacggccgg aaacgaatgc tcgctagtgc gggcgagctg 3660
cagaaaggta acgagctggc actgccctct aaatacgtta atttcttgta tctggccagc 3720
cactatgaaa agctcaaagg gtctcccgaa gataatgagc agaagcagct gttcgtggaa 3780
caacacaaac actaccttga tgagatcatc gagcaaataa gcgaattctc caaaagagtg 3840
atcctcgccg acgctaacct cgataaggtg ctttctgctt acaataagca cagggataag 3900
cccatcaggg agcaggcaga aaacattatc cacttgttta ctctgaccaa cttgggcgcg 3960
cctgcagcct tcaagtactt cgacaccacc atagacagaa agcggtacac ctctacaaag 4020
gaggtcctgg acgccacact gattcatcag tcaattacgg ggctctatga aacaagaatc 4080
gacctctctc agctcggtgg agac 4104
<210> 66
<211> 4104
<212> DNA
<213> Artificial Sequence
<220>
<223> dead Cas9 (dCas9) nucleic acid
<400> 66
atggacaaga agtactccat tgggctcgct atcggcacaa acagcgtcgg ctgggccgtc 60
attacggacg agtacaaggt gccgagcaaa aaattcaaag ttctgggcaa taccgatcgc 120
cacagcataa agaagaacct cattggcgcc ctcctgttcg actccgggga gacggccgaa 180
gccacgcggc tcaaaagaac agcacggcgc agatataccc gcagaaagaa tcggatctgc 240
tacctgcagg agatctttag taatgagatg gctaaggtgg atgactcttt cttccatagg 300
ctggaggagt cctttttggt ggaggaggat aaaaagcacg agcgccaccc aatctttggc 360
aatatcgtgg acgaggtggc gtaccatgaa aagtacccaa ccatatatca tctgaggaag 420
aagcttgtag acagtactga taaggctgac ttgcggttga tctatctcgc gctggcgcat 480
atgatcaaat ttcggggaca cttcctcatc gagggggacc tgaacccaga caacagcgat 540
gtcgacaaac tctttatcca actggttcag acttacaatc agcttttcga agagaacccg 600
atcaacgcat ccggagttga cgccaaagca atcctgagcg ctaggctgtc caaatcccgg 660
cggctcgaaa acctcatcgc acagctccct ggggagaaga agaacggcct gtttggtaat 720
cttatcgccc tgtcactcgg gctgaccccc aactttaaat ctaacttcga cctggccgaa 780
gatgccaagc ttcaactgag caaagacacc tacgatgatg atctcgacaa tctgctggcc 840
cagatcggcg accagtacgc agaccttttt ttggcggcaa agaacctgtc agacgccatt 900
ctgctgagtg atattctgcg agtgaacacg gagatcacca aagctccgct gagcgctagt 960
atgatcaagc gctatgatga gcaccaccaa gacttgactt tgctgaaggc ccttgtcaga 1020
cagcaactgc ctgagaagta caaggaaatt ttcttcgatc agtctaaaaa tggctacgcc 1080
ggatacattg acggcggagc aagccaggag gaattttaca aatttattaa gcccatcttg 1140
gaaaaaatgg acggcaccga ggagctgctg gtaaagctta acagagaaga tctgttgcgc 1200
aaacagcgca ctttcgacaa tggaagcatc ccccaccaga ttcacctggg cgaactgcac 1260
gctatcctca ggcggcaaga ggatttctac ccctttttga aagataacag ggaaaagatt 1320
gagaaaatcc tcacatttcg gataccctac tatgtaggcc ccctcgcccg gggaaattcc 1380
agattcgcgt ggatgactcg caaatcagaa gagaccatca ctccctggaa cttcgaggaa 1440
gtcgtggata agggggcctc tgcccagtcc ttcatcgaaa ggatgactaa ctttgataaa 1500
aatctgccta acgaaaaggt gcttcctaaa cactctctgc tgtacgagta cttcacagtt 1560
tataacgagc tcaccaaggt caaatacgtc acagaaggga tgagaaagcc agcattcctg 1620
tctggagagc agaagaaagc tatcgtggac ctcctcttca agacgaaccg gaaagttacc 1680
gtgaaacagc tcaaagaaga ctatttcaaa aagattgaat gtttcgactc tgttgaaatc 1740
agcggagtgg aggatcgctt caacgcatcc ctgggaacgt atcacgatct cctgaaaatc 1800
attaaagaca aggacttcct ggacaatgag gagaacgagg acattcttga ggacattgtc 1860
ctcaccctta cgttgtttga agatagggag atgattgaag aacgcttgaa aacttacgct 1920
catctcttcg acgacaaagt catgaaacag ctcaagaggc gccgatatac aggatggggg 1980
cggctgtcaa gaaaactgat caatgggatc cgagacaagc agagtggaaa gacaatcctg 2040
gattttctta agtccgatgg atttgccaac cggaacttca tgcagttgat ccatgatgac 2100
tctctcacct ttaaggagga catccagaaa gcacaagttt ctggccaggg ggacagtctt 2160
cacgagcaca tcgctaatct tgcaggtagc ccagctatca aaaagggaat actgcagacc 2220
gttaaggtcg tggatgaact cgtcaaagta atgggaaggc ataagcccga gaatatcgtt 2280
atcgagatgg cccgagagaa ccaaactacc cagaagggac agaagaacag tagggaaagg 2340
atgaagagga ttgaagaggg tataaaagaa ctggggtccc aaatccttaa ggaacaccca 2400
gttgaaaaca cccagcttca gaatgagaag ctctacctgt actacctgca gaacggcagg 2460
gacatgtacg tggatcagga actggacatc aatcggctct ccgactacga cgtggctgct 2520
atcgtgcccc agtcttttct caaagatgat tctattgata ataaagtgtt gacaagatcc 2580
gataaagcta gagggaagag tgataacgtc ccctcagaag aagttgtcaa gaaaatgaaa 2640
aattattggc ggcagctgct gaacgccaaa ctgatcacac aacggaagtt cgataatctg 2700
actaaggctg aacgaggtgg cctgtctgag ttggataaag ccggcttcat caaaaggcag 2760
cttgttgaga cacgccagat caccaagcac gtggcccaaa ttctcgattc acgcatgaac 2820
accaagtacg atgaaaatga caaactgatt cgagaggtga aagttattac tctgaagtct 2880
aagctggtct cagatttcag aaaggacttt cagttttata aggtgagaga gatcaacaat 2940
taccaccatg cgcatgatgc ctacctgaat gcagtggtag gcactgcact tatcaaaaaa 3000
tatcccaagc ttgaatctga atttgtttac ggagactata aagtgtacga tgttaggaaa 3060
atgatcgcaa agtctgagca ggaaataggc aaggccaccg ctaagtactt cttttacagc 3120
aatattatga attttttcaa gaccgagatt acactggcca atggagagat tcggaagcga 3180
ccacttatcg aaacaaacgg agaaacagga gaaatcgtgt gggacaaggg tagggatttc 3240
gcgacagtcc ggaaggtcct gtccatgccg caggtgaaca tcgttaaaaa gaccgaagta 3300
cagaccggag gcttctccaa ggaaagtatc ctcccgaaaa ggaacagcga caagctgatc 3360
gcacgcaaaa aagattggga ccccaagaaa tacggcggat tcgattctcc tacagtcgct 3420
tacagtgtac tggttgtggc caaagtggag aaagggaagt ctaaaaaact caaaagcgtc 3480
aaggaactgc tgggcatcac aatcatggag cgatcaagct tcgaaaaaaa ccccatcgac 3540
tttctcgagg cgaaaggata taaagaggtc aaaaaagacc tcatcattaa gcttcccaag 3600
tactctctct ttgagcttga aaacggccgg aaacgaatgc tcgctagtgc gggcgagctg 3660
cagaaaggta acgagctggc actgccctct aaatacgtta atttcttgta tctggccagc 3720
cactatgaaa agctcaaagg gtctcccgaa gataatgagc agaagcagct gttcgtggaa 3780
caacacaaac actaccttga tgagatcatc gagcaaataa gcgaattctc caaaagagtg 3840
atcctcgccg acgctaacct cgataaggtg ctttctgctt acaataagca cagggataag 3900
cccatcaggg agcaggcaga aaacattatc cacttgttta ctctgaccaa cttgggcgcg 3960
cctgcagcct tcaagtactt cgacaccacc atagacagaa agcggtacac ctctacaaag 4020
gaggtcctgg acgccacact gattcatcag tcaattacgg ggctctatga aacaagaatc 4080
gacctctctc agctcggtgg agac 4104
<210> 67
<211> 1782
<212> DNA
<213> Artificial Sequence
<220>
<223> hyperactive PiggyBac (PB) transposase nucleic acid
<400> 67
atgggcagca gcctggacga cgagcacatc ctgagcgccc tgctgcagag cgacgacgag 60
ctggtcggcg aggacagcga cagcgaggtg agcgaccacg tgagcgagga cgacgtgcag 120
tccgacaccg aggaggcctt catcgacgag gtgcacgagg tgcagcctac cagcagcggc 180
tccgagatcc tggacgagca gaacgtgatc gagcagcccg gcagctccct ggccagcaac 240
aggatcctga ccctgcccca gaggaccatc aggggcaaga acaagcactg ctggtccacc 300
tccaagccca ccaggcggag cagggtgtcc gccctgaaca tcgtgagaag ccagaggggc 360
cccaccagga tgtgcaggaa catctacgac cccctgctgt gcttcaagct gttcttcacc 420
gacgagatca tcagcgagat cgtgaagtgg accaacgccg agatcagcct gaagaggcgg 480
gagagcatga cctccgccac cttcagggac accaacgagg acgagatcta cgccttcttc 540
ggcatcctgg tgatgaccgc cgtgaggaag gacaaccaca tgagcaccga cgacctgttc 600
gacagatccc tgagcatggt gtacgtgagc gtgatgagca gggacagatt cgacttcctg 660
atcagatgcc tgaggatgga cgacaagagc atcaggccca ccctgcggga gaacgacgtg 720
ttcacccccg tgagaaagat ctgggacctg ttcatccacc agtgcatcca gaactacacc 780
cctggcgccc acctgaccat cgacgagcag ctgctgggct tcaggggcag gtgccccttc 840
agggtctata tccccaacaa gcccagcaag tacggcatca agatcctgat gatgtgcgac 900
agcggcacca agtacatgat caacggcatg ccctacctgg gcaggggcac ccagaccaac 960
ggcgtgcccc tgggcgagta ctacgtgaag gagctgtcca agcccgtcca cggcagctgc 1020
agaaacatca cctgcgacaa ctggttcacc agcatccccc tggccaagaa cctgctgcag 1080
gagccctaca agctgaccat cgtgggcacc gtgagaagca acaagagaga gatccccgag 1140
gtcctgaaga acagcaggtc caggcccgtg ggcaccagca tgttctgctt cgacggcccc 1200
ctgaccctgg tgtcctacaa gcccaagccc gccaagatgg tgtacctgct gtccagctgc 1260
gacgaggacg ccagcatcaa cgagagcacc ggcaagcccc agatggtgat gtactacaac 1320
cagaccaagg gcggcgtgga caccctggac cagatgtgca gcgtgatgac ctgcagcaga 1380
aagaccaaca ggtggcccat ggccctgctg tacggcatga tcaacatcgc ctgcatcaac 1440
agcttcatca tctacagcca caacgtgagc agcaagggcg agaaggtgca gagccggaaa 1500
aagttcatgc ggaacctgta catgggcctg acctccagct tcatgaggaa gaggctggag 1560
gcccccaccc tgaagagata cctgagggac aacatcagca acatcctgcc caaagaggtg 1620
cccggcacca gcgacgacag caccgaggag cccgtgatga agaagaggac ctactgcacc 1680
tactgtccca gcaagatcag aagaaaggcc agcgccagct gcaagaagtg taagaaggtc 1740
atctgccggg agcacaacat cgacatgtgc cagagctgtt tc 1782
<210> 68
<211> 1020
<212> DNA
<213> Artificial Sequence
<220>
<223> hyperactive Sleeping Beauty (SB100) transposase nucleic acid
<400> 68
atgggaaaat caaaagaaat cagccaagac ctcagaaaaa gaattgtaga cctccacaag 60
tctggttcat ccttgggagc aatttccaaa cgcctggcgg taccacgttc atctgtacaa 120
acaatagtac gcaagtataa acaccatggg accacgcagc cgtcataccg ctcaggaagg 180
agacgcgttc tgtctcctag agatgaacgt actttggtgc gaaaagtgca aatcaatccc 240
agaacaacag caaaggacct tgtgaagatg ctggaggaaa caggtacaaa agtatctata 300
tccacagtaa aacgagtcct atatcgacat aacctgaaag gccactcagc aaggaagaag 360
ccactgctcc aaaaccgaca taagaaagcc agactacggt ttgcaactgc acatggggac 420
aaagatcgta ctttttggag aaatgtcctc tggtctgatg aaacaaaaat agaactgttt 480
ggccataatg accatcgtta tgtttggagg aagaaggggg aggcttgcaa gccgaagaac 540
accatcccaa ccgtgaagca cgggggtggc agcatcatgt tgtgggggtg ctttgctgca 600
ggagggactg gtgcacttca caaaatagat ggcatcatgg acgccgtgca gtatgtggat 660
atattgaagc aacatctcaa gacatcagtc aggaagttaa agcttggtcg caaatgggtc 720
ttccaacacg acaatgaccc caagcatact tccaaagttg tggcaaaatg gcttaaggac 780
aacaaagtca aggtattgga gtggccatca caaagccctg acctcaatcc tatagaaaat 840
ttgtgggcag aactgaaaaa gcgtgtgcga gcaaggaggc ctacaaacct gactcagtta 900
caccagctct gtcaggagga atgggccaaa attcacccaa attattgtgg gaagcttgtg 960
gaaggctacc cgaaacgttt gacccaagtt aaacaattta aaggcaatgc taccaaatac 1020
<210> 69
<211> 1368
<212> PRT
<213> Artificial Sequence
<220>
<223> human Cas9 (hCas9) amino acid
<400> 69
Met Asp Lys Lys Tyr Ser Ile Gly Leu Asp Ile Gly Thr Asn Ser Val
1 5 10 15
Gly Trp Ala Val Ile Thr Asp Glu Tyr Lys Val Pro Ser Lys Lys Phe
20 25 30
Lys Val Leu Gly Asn Thr Asp Arg His Ser Ile Lys Lys Asn Leu Ile
35 40 45
Gly Ala Leu Leu Phe Asp Ser Gly Glu Thr Ala Glu Ala Thr Arg Leu
50 55 60
Lys Arg Thr Ala Arg Arg Arg Tyr Thr Arg Arg Lys Asn Arg Ile Cys
65 70 75 80
Tyr Leu Gln Glu Ile Phe Ser Asn Glu Met Ala Lys Val Asp Asp Ser
85 90 95
Phe Phe His Arg Leu Glu Glu Ser Phe Leu Val Glu Glu Asp Lys Lys
100 105 110
His Glu Arg His Pro Ile Phe Gly Asn Ile Val Asp Glu Val Ala Tyr
115 120 125
His Glu Lys Tyr Pro Thr Ile Tyr His Leu Arg Lys Lys Leu Val Asp
130 135 140
Ser Thr Asp Lys Ala Asp Leu Arg Leu Ile Tyr Leu Ala Leu Ala His
145 150 155 160
Met Ile Lys Phe Arg Gly His Phe Leu Ile Glu Gly Asp Leu Asn Pro
165 170 175
Asp Asn Ser Asp Val Asp Lys Leu Phe Ile Gln Leu Val Gln Thr Tyr
180 185 190
Asn Gln Leu Phe Glu Glu Asn Pro Ile Asn Ala Ser Gly Val Asp Ala
195 200 205
Lys Ala Ile Leu Ser Ala Arg Leu Ser Lys Ser Arg Arg Leu Glu Asn
210 215 220
Leu Ile Ala Gln Leu Pro Gly Glu Lys Lys Asn Gly Leu Phe Gly Asn
225 230 235 240
Leu Ile Ala Leu Ser Leu Gly Leu Thr Pro Asn Phe Lys Ser Asn Phe
245 250 255
Asp Leu Ala Glu Asp Ala Lys Leu Gln Leu Ser Lys Asp Thr Tyr Asp
260 265 270
Asp Asp Leu Asp Asn Leu Leu Ala Gln Ile Gly Asp Gln Tyr Ala Asp
275 280 285
Leu Phe Leu Ala Ala Lys Asn Leu Ser Asp Ala Ile Leu Leu Ser Asp
290 295 300
Ile Leu Arg Val Asn Thr Glu Ile Thr Lys Ala Pro Leu Ser Ala Ser
305 310 315 320
Met Ile Lys Arg Tyr Asp Glu His His Gln Asp Leu Thr Leu Leu Lys
325 330 335
Ala Leu Val Arg Gln Gln Leu Pro Glu Lys Tyr Lys Glu Ile Phe Phe
340 345 350
Asp Gln Ser Lys Asn Gly Tyr Ala Gly Tyr Ile Asp Gly Gly Ala Ser
355 360 365
Gln Glu Glu Phe Tyr Lys Phe Ile Lys Pro Ile Leu Glu Lys Met Asp
370 375 380
Gly Thr Glu Glu Leu Leu Val Lys Leu Asn Arg Glu Asp Leu Leu Arg
385 390 395 400
Lys Gln Arg Thr Phe Asp Asn Gly Ser Ile Pro His Gln Ile His Leu
405 410 415
Gly Glu Leu His Ala Ile Leu Arg Arg Gln Glu Asp Phe Tyr Pro Phe
420 425 430
Leu Lys Asp Asn Arg Glu Lys Ile Glu Lys Ile Leu Thr Phe Arg Ile
435 440 445
Pro Tyr Tyr Val Gly Pro Leu Ala Arg Gly Asn Ser Arg Phe Ala Trp
450 455 460
Met Thr Arg Lys Ser Glu Glu Thr Ile Thr Pro Trp Asn Phe Glu Glu
465 470 475 480
Val Val Asp Lys Gly Ala Ser Ala Gln Ser Phe Ile Glu Arg Met Thr
485 490 495
Asn Phe Asp Lys Asn Leu Pro Asn Glu Lys Val Leu Pro Lys His Ser
500 505 510
Leu Leu Tyr Glu Tyr Phe Thr Val Tyr Asn Glu Leu Thr Lys Val Lys
515 520 525
Tyr Val Thr Glu Gly Met Arg Lys Pro Ala Phe Leu Ser Gly Glu Gln
530 535 540
Lys Lys Ala Ile Val Asp Leu Leu Phe Lys Thr Asn Arg Lys Val Thr
545 550 555 560
Val Lys Gln Leu Lys Glu Asp Tyr Phe Lys Lys Ile Glu Cys Phe Asp
565 570 575
Ser Val Glu Ile Ser Gly Val Glu Asp Arg Phe Asn Ala Ser Leu Gly
580 585 590
Thr Tyr His Asp Leu Leu Lys Ile Ile Lys Asp Lys Asp Phe Leu Asp
595 600 605
Asn Glu Glu Asn Glu Asp Ile Leu Glu Asp Ile Val Leu Thr Leu Thr
610 615 620
Leu Phe Glu Asp Arg Glu Met Ile Glu Glu Arg Leu Lys Thr Tyr Ala
625 630 635 640
His Leu Phe Asp Asp Lys Val Met Lys Gln Leu Lys Arg Arg Arg Tyr
645 650 655
Thr Gly Trp Gly Arg Leu Ser Arg Lys Leu Ile Asn Gly Ile Arg Asp
660 665 670
Lys Gln Ser Gly Lys Thr Ile Leu Asp Phe Leu Lys Ser Asp Gly Phe
675 680 685
Ala Asn Arg Asn Phe Met Gln Leu Ile His Asp Asp Ser Leu Thr Phe
690 695 700
Lys Glu Asp Ile Gln Lys Ala Gln Val Ser Gly Gln Gly Asp Ser Leu
705 710 715 720
His Glu His Ile Ala Asn Leu Ala Gly Ser Pro Ala Ile Lys Lys Gly
725 730 735
Ile Leu Gln Thr Val Lys Val Val Asp Glu Leu Val Lys Val Met Gly
740 745 750
Arg His Lys Pro Glu Asn Ile Val Ile Glu Met Ala Arg Glu Asn Gln
755 760 765
Thr Thr Gln Lys Gly Gln Lys Asn Ser Arg Glu Arg Met Lys Arg Ile
770 775 780
Glu Glu Gly Ile Lys Glu Leu Gly Ser Gln Ile Leu Lys Glu His Pro
785 790 795 800
Val Glu Asn Thr Gln Leu Gln Asn Glu Lys Leu Tyr Leu Tyr Tyr Leu
805 810 815
Gln Asn Gly Arg Asp Met Tyr Val Asp Gln Glu Leu Asp Ile Asn Arg
820 825 830
Leu Ser Asp Tyr Asp Val Asp His Ile Val Pro Gln Ser Phe Leu Lys
835 840 845
Asp Asp Ser Ile Asp Asn Lys Val Leu Thr Arg Ser Asp Lys Asn Arg
850 855 860
Gly Lys Ser Asp Asn Val Pro Ser Glu Glu Val Val Lys Lys Met Lys
865 870 875 880
Asn Tyr Trp Arg Gln Leu Leu Asn Ala Lys Leu Ile Thr Gln Arg Lys
885 890 895
Phe Asp Asn Leu Thr Lys Ala Glu Arg Gly Gly Leu Ser Glu Leu Asp
900 905 910
Lys Ala Gly Phe Ile Lys Arg Gln Leu Val Glu Thr Arg Gln Ile Thr
915 920 925
Lys His Val Ala Gln Ile Leu Asp Ser Arg Met Asn Thr Lys Tyr Asp
930 935 940
Glu Asn Asp Lys Leu Ile Arg Glu Val Lys Val Ile Thr Leu Lys Ser
945 950 955 960
Lys Leu Val Ser Asp Phe Arg Lys Asp Phe Gln Phe Tyr Lys Val Arg
965 970 975
Glu Ile Asn Asn Tyr His His Ala His Asp Ala Tyr Leu Asn Ala Val
980 985 990
Val Gly Thr Ala Leu Ile Lys Lys Tyr Pro Lys Leu Glu Ser Glu Phe
995 1000 1005
Val Tyr Gly Asp Tyr Lys Val Tyr Asp Val Arg Lys Met Ile Ala
1010 1015 1020
Lys Ser Glu Gln Glu Ile Gly Lys Ala Thr Ala Lys Tyr Phe Phe
1025 1030 1035
Tyr Ser Asn Ile Met Asn Phe Phe Lys Thr Glu Ile Thr Leu Ala
1040 1045 1050
Asn Gly Glu Ile Arg Lys Arg Pro Leu Ile Glu Thr Asn Gly Glu
1055 1060 1065
Thr Gly Glu Ile Val Trp Asp Lys Gly Arg Asp Phe Ala Thr Val
1070 1075 1080
Arg Lys Val Leu Ser Met Pro Gln Val Asn Ile Val Lys Lys Thr
1085 1090 1095
Glu Val Gln Thr Gly Gly Phe Ser Lys Glu Ser Ile Leu Pro Lys
1100 1105 1110
Arg Asn Ser Asp Lys Leu Ile Ala Arg Lys Lys Asp Trp Asp Pro
1115 1120 1125
Lys Lys Tyr Gly Gly Phe Asp Ser Pro Thr Val Ala Tyr Ser Val
1130 1135 1140
Leu Val Val Ala Lys Val Glu Lys Gly Lys Ser Lys Lys Leu Lys
1145 1150 1155
Ser Val Lys Glu Leu Leu Gly Ile Thr Ile Met Glu Arg Ser Ser
1160 1165 1170
Phe Glu Lys Asn Pro Ile Asp Phe Leu Glu Ala Lys Gly Tyr Lys
1175 1180 1185
Glu Val Lys Lys Asp Leu Ile Ile Lys Leu Pro Lys Tyr Ser Leu
1190 1195 1200
Phe Glu Leu Glu Asn Gly Arg Lys Arg Met Leu Ala Ser Ala Gly
1205 1210 1215
Glu Leu Gln Lys Gly Asn Glu Leu Ala Leu Pro Ser Lys Tyr Val
1220 1225 1230
Asn Phe Leu Tyr Leu Ala Ser His Tyr Glu Lys Leu Lys Gly Ser
1235 1240 1245
Pro Glu Asp Asn Glu Gln Lys Gln Leu Phe Val Glu Gln His Lys
1250 1255 1260
His Tyr Leu Asp Glu Ile Ile Glu Gln Ile Ser Glu Phe Ser Lys
1265 1270 1275
Arg Val Ile Leu Ala Asp Ala Asn Leu Asp Lys Val Leu Ser Ala
1280 1285 1290
Tyr Asn Lys His Arg Asp Lys Pro Ile Arg Glu Gln Ala Glu Asn
1295 1300 1305
Ile Ile His Leu Phe Thr Leu Thr Asn Leu Gly Ala Pro Ala Ala
1310 1315 1320
Phe Lys Tyr Phe Asp Thr Thr Ile Asp Arg Lys Arg Tyr Thr Ser
1325 1330 1335
Thr Lys Glu Val Leu Asp Ala Thr Leu Ile His Gln Ser Ile Thr
1340 1345 1350
Gly Leu Tyr Glu Thr Arg Ile Asp Leu Ser Gln Leu Gly Gly Asp
1355 1360 1365
<210> 70
<211> 1368
<212> PRT
<213> Artificial Sequence
<220>
<223> nickase Cas9 (nCas9) amino acid
<400> 70
Met Asp Lys Lys Tyr Ser Ile Gly Leu Ala Ile Gly Thr Asn Ser Val
1 5 10 15
Gly Trp Ala Val Ile Thr Asp Glu Tyr Lys Val Pro Ser Lys Lys Phe
20 25 30
Lys Val Leu Gly Asn Thr Asp Arg His Ser Ile Lys Lys Asn Leu Ile
35 40 45
Gly Ala Leu Leu Phe Asp Ser Gly Glu Thr Ala Glu Ala Thr Arg Leu
50 55 60
Lys Arg Thr Ala Arg Arg Arg Tyr Thr Arg Arg Lys Asn Arg Ile Cys
65 70 75 80
Tyr Leu Gln Glu Ile Phe Ser Asn Glu Met Ala Lys Val Asp Asp Ser
85 90 95
Phe Phe His Arg Leu Glu Glu Ser Phe Leu Val Glu Glu Asp Lys Lys
100 105 110
His Glu Arg His Pro Ile Phe Gly Asn Ile Val Asp Glu Val Ala Tyr
115 120 125
His Glu Lys Tyr Pro Thr Ile Tyr His Leu Arg Lys Lys Leu Val Asp
130 135 140
Ser Thr Asp Lys Ala Asp Leu Arg Leu Ile Tyr Leu Ala Leu Ala His
145 150 155 160
Met Ile Lys Phe Arg Gly His Phe Leu Ile Glu Gly Asp Leu Asn Pro
165 170 175
Asp Asn Ser Asp Val Asp Lys Leu Phe Ile Gln Leu Val Gln Thr Tyr
180 185 190
Asn Gln Leu Phe Glu Glu Asn Pro Ile Asn Ala Ser Gly Val Asp Ala
195 200 205
Lys Ala Ile Leu Ser Ala Arg Leu Ser Lys Ser Arg Arg Leu Glu Asn
210 215 220
Leu Ile Ala Gln Leu Pro Gly Glu Lys Lys Asn Gly Leu Phe Gly Asn
225 230 235 240
Leu Ile Ala Leu Ser Leu Gly Leu Thr Pro Asn Phe Lys Ser Asn Phe
245 250 255
Asp Leu Ala Glu Asp Ala Lys Leu Gln Leu Ser Lys Asp Thr Tyr Asp
260 265 270
Asp Asp Leu Asp Asn Leu Leu Ala Gln Ile Gly Asp Gln Tyr Ala Asp
275 280 285
Leu Phe Leu Ala Ala Lys Asn Leu Ser Asp Ala Ile Leu Leu Ser Asp
290 295 300
Ile Leu Arg Val Asn Thr Glu Ile Thr Lys Ala Pro Leu Ser Ala Ser
305 310 315 320
Met Ile Lys Arg Tyr Asp Glu His His Gln Asp Leu Thr Leu Leu Lys
325 330 335
Ala Leu Val Arg Gln Gln Leu Pro Glu Lys Tyr Lys Glu Ile Phe Phe
340 345 350
Asp Gln Ser Lys Asn Gly Tyr Ala Gly Tyr Ile Asp Gly Gly Ala Ser
355 360 365
Gln Glu Glu Phe Tyr Lys Phe Ile Lys Pro Ile Leu Glu Lys Met Asp
370 375 380
Gly Thr Glu Glu Leu Leu Val Lys Leu Asn Arg Glu Asp Leu Leu Arg
385 390 395 400
Lys Gln Arg Thr Phe Asp Asn Gly Ser Ile Pro His Gln Ile His Leu
405 410 415
Gly Glu Leu His Ala Ile Leu Arg Arg Gln Glu Asp Phe Tyr Pro Phe
420 425 430
Leu Lys Asp Asn Arg Glu Lys Ile Glu Lys Ile Leu Thr Phe Arg Ile
435 440 445
Pro Tyr Tyr Val Gly Pro Leu Ala Arg Gly Asn Ser Arg Phe Ala Trp
450 455 460
Met Thr Arg Lys Ser Glu Glu Thr Ile Thr Pro Trp Asn Phe Glu Glu
465 470 475 480
Val Val Asp Lys Gly Ala Ser Ala Gln Ser Phe Ile Glu Arg Met Thr
485 490 495
Asn Phe Asp Lys Asn Leu Pro Asn Glu Lys Val Leu Pro Lys His Ser
500 505 510
Leu Leu Tyr Glu Tyr Phe Thr Val Tyr Asn Glu Leu Thr Lys Val Lys
515 520 525
Tyr Val Thr Glu Gly Met Arg Lys Pro Ala Phe Leu Ser Gly Glu Gln
530 535 540
Lys Lys Ala Ile Val Asp Leu Leu Phe Lys Thr Asn Arg Lys Val Thr
545 550 555 560
Val Lys Gln Leu Lys Glu Asp Tyr Phe Lys Lys Ile Glu Cys Phe Asp
565 570 575
Ser Val Glu Ile Ser Gly Val Glu Asp Arg Phe Asn Ala Ser Leu Gly
580 585 590
Thr Tyr His Asp Leu Leu Lys Ile Ile Lys Asp Lys Asp Phe Leu Asp
595 600 605
Asn Glu Glu Asn Glu Asp Ile Leu Glu Asp Ile Val Leu Thr Leu Thr
610 615 620
Leu Phe Glu Asp Arg Glu Met Ile Glu Glu Arg Leu Lys Thr Tyr Ala
625 630 635 640
His Leu Phe Asp Asp Lys Val Met Lys Gln Leu Lys Arg Arg Arg Tyr
645 650 655
Thr Gly Trp Gly Arg Leu Ser Arg Lys Leu Ile Asn Gly Ile Arg Asp
660 665 670
Lys Gln Ser Gly Lys Thr Ile Leu Asp Phe Leu Lys Ser Asp Gly Phe
675 680 685
Ala Asn Arg Asn Phe Met Gln Leu Ile His Asp Asp Ser Leu Thr Phe
690 695 700
Lys Glu Asp Ile Gln Lys Ala Gln Val Ser Gly Gln Gly Asp Ser Leu
705 710 715 720
His Glu His Ile Ala Asn Leu Ala Gly Ser Pro Ala Ile Lys Lys Gly
725 730 735
Ile Leu Gln Thr Val Lys Val Val Asp Glu Leu Val Lys Val Met Gly
740 745 750
Arg His Lys Pro Glu Asn Ile Val Ile Glu Met Ala Arg Glu Asn Gln
755 760 765
Thr Thr Gln Lys Gly Gln Lys Asn Ser Arg Glu Arg Met Lys Arg Ile
770 775 780
Glu Glu Gly Ile Lys Glu Leu Gly Ser Gln Ile Leu Lys Glu His Pro
785 790 795 800
Val Glu Asn Thr Gln Leu Gln Asn Glu Lys Leu Tyr Leu Tyr Tyr Leu
805 810 815
Gln Asn Gly Arg Asp Met Tyr Val Asp Gln Glu Leu Asp Ile Asn Arg
820 825 830
Leu Ser Asp Tyr Asp Val Asp His Ile Val Pro Gln Ser Phe Leu Lys
835 840 845
Asp Asp Ser Ile Asp Asn Lys Val Leu Thr Arg Ser Asp Lys Asn Arg
850 855 860
Gly Lys Ser Asp Asn Val Pro Ser Glu Glu Val Val Lys Lys Met Lys
865 870 875 880
Asn Tyr Trp Arg Gln Leu Leu Asn Ala Lys Leu Ile Thr Gln Arg Lys
885 890 895
Phe Asp Asn Leu Thr Lys Ala Glu Arg Gly Gly Leu Ser Glu Leu Asp
900 905 910
Lys Ala Gly Phe Ile Lys Arg Gln Leu Val Glu Thr Arg Gln Ile Thr
915 920 925
Lys His Val Ala Gln Ile Leu Asp Ser Arg Met Asn Thr Lys Tyr Asp
930 935 940
Glu Asn Asp Lys Leu Ile Arg Glu Val Lys Val Ile Thr Leu Lys Ser
945 950 955 960
Lys Leu Val Ser Asp Phe Arg Lys Asp Phe Gln Phe Tyr Lys Val Arg
965 970 975
Glu Ile Asn Asn Tyr His His Ala His Asp Ala Tyr Leu Asn Ala Val
980 985 990
Val Gly Thr Ala Leu Ile Lys Lys Tyr Pro Lys Leu Glu Ser Glu Phe
995 1000 1005
Val Tyr Gly Asp Tyr Lys Val Tyr Asp Val Arg Lys Met Ile Ala
1010 1015 1020
Lys Ser Glu Gln Glu Ile Gly Lys Ala Thr Ala Lys Tyr Phe Phe
1025 1030 1035
Tyr Ser Asn Ile Met Asn Phe Phe Lys Thr Glu Ile Thr Leu Ala
1040 1045 1050
Asn Gly Glu Ile Arg Lys Arg Pro Leu Ile Glu Thr Asn Gly Glu
1055 1060 1065
Thr Gly Glu Ile Val Trp Asp Lys Gly Arg Asp Phe Ala Thr Val
1070 1075 1080
Arg Lys Val Leu Ser Met Pro Gln Val Asn Ile Val Lys Lys Thr
1085 1090 1095
Glu Val Gln Thr Gly Gly Phe Ser Lys Glu Ser Ile Leu Pro Lys
1100 1105 1110
Arg Asn Ser Asp Lys Leu Ile Ala Arg Lys Lys Asp Trp Asp Pro
1115 1120 1125
Lys Lys Tyr Gly Gly Phe Asp Ser Pro Thr Val Ala Tyr Ser Val
1130 1135 1140
Leu Val Val Ala Lys Val Glu Lys Gly Lys Ser Lys Lys Leu Lys
1145 1150 1155
Ser Val Lys Glu Leu Leu Gly Ile Thr Ile Met Glu Arg Ser Ser
1160 1165 1170
Phe Glu Lys Asn Pro Ile Asp Phe Leu Glu Ala Lys Gly Tyr Lys
1175 1180 1185
Glu Val Lys Lys Asp Leu Ile Ile Lys Leu Pro Lys Tyr Ser Leu
1190 1195 1200
Phe Glu Leu Glu Asn Gly Arg Lys Arg Met Leu Ala Ser Ala Gly
1205 1210 1215
Glu Leu Gln Lys Gly Asn Glu Leu Ala Leu Pro Ser Lys Tyr Val
1220 1225 1230
Asn Phe Leu Tyr Leu Ala Ser His Tyr Glu Lys Leu Lys Gly Ser
1235 1240 1245
Pro Glu Asp Asn Glu Gln Lys Gln Leu Phe Val Glu Gln His Lys
1250 1255 1260
His Tyr Leu Asp Glu Ile Ile Glu Gln Ile Ser Glu Phe Ser Lys
1265 1270 1275
Arg Val Ile Leu Ala Asp Ala Asn Leu Asp Lys Val Leu Ser Ala
1280 1285 1290
Tyr Asn Lys His Arg Asp Lys Pro Ile Arg Glu Gln Ala Glu Asn
1295 1300 1305
Ile Ile His Leu Phe Thr Leu Thr Asn Leu Gly Ala Pro Ala Ala
1310 1315 1320
Phe Lys Tyr Phe Asp Thr Thr Ile Asp Arg Lys Arg Tyr Thr Ser
1325 1330 1335
Thr Lys Glu Val Leu Asp Ala Thr Leu Ile His Gln Ser Ile Thr
1340 1345 1350
Gly Leu Tyr Glu Thr Arg Ile Asp Leu Ser Gln Leu Gly Gly Asp
1355 1360 1365
<210> 71
<211> 1368
<212> PRT
<213> Artificial Sequence
<220>
<223> dead Cas9 (dCas9) amino acid
<400> 71
Met Asp Lys Lys Tyr Ser Ile Gly Leu Ala Ile Gly Thr Asn Ser Val
1 5 10 15
Gly Trp Ala Val Ile Thr Asp Glu Tyr Lys Val Pro Ser Lys Lys Phe
20 25 30
Lys Val Leu Gly Asn Thr Asp Arg His Ser Ile Lys Lys Asn Leu Ile
35 40 45
Gly Ala Leu Leu Phe Asp Ser Gly Glu Thr Ala Glu Ala Thr Arg Leu
50 55 60
Lys Arg Thr Ala Arg Arg Arg Tyr Thr Arg Arg Lys Asn Arg Ile Cys
65 70 75 80
Tyr Leu Gln Glu Ile Phe Ser Asn Glu Met Ala Lys Val Asp Asp Ser
85 90 95
Phe Phe His Arg Leu Glu Glu Ser Phe Leu Val Glu Glu Asp Lys Lys
100 105 110
His Glu Arg His Pro Ile Phe Gly Asn Ile Val Asp Glu Val Ala Tyr
115 120 125
His Glu Lys Tyr Pro Thr Ile Tyr His Leu Arg Lys Lys Leu Val Asp
130 135 140
Ser Thr Asp Lys Ala Asp Leu Arg Leu Ile Tyr Leu Ala Leu Ala His
145 150 155 160
Met Ile Lys Phe Arg Gly His Phe Leu Ile Glu Gly Asp Leu Asn Pro
165 170 175
Asp Asn Ser Asp Val Asp Lys Leu Phe Ile Gln Leu Val Gln Thr Tyr
180 185 190
Asn Gln Leu Phe Glu Glu Asn Pro Ile Asn Ala Ser Gly Val Asp Ala
195 200 205
Lys Ala Ile Leu Ser Ala Arg Leu Ser Lys Ser Arg Arg Leu Glu Asn
210 215 220
Leu Ile Ala Gln Leu Pro Gly Glu Lys Lys Asn Gly Leu Phe Gly Asn
225 230 235 240
Leu Ile Ala Leu Ser Leu Gly Leu Thr Pro Asn Phe Lys Ser Asn Phe
245 250 255
Asp Leu Ala Glu Asp Ala Lys Leu Gln Leu Ser Lys Asp Thr Tyr Asp
260 265 270
Asp Asp Leu Asp Asn Leu Leu Ala Gln Ile Gly Asp Gln Tyr Ala Asp
275 280 285
Leu Phe Leu Ala Ala Lys Asn Leu Ser Asp Ala Ile Leu Leu Ser Asp
290 295 300
Ile Leu Arg Val Asn Thr Glu Ile Thr Lys Ala Pro Leu Ser Ala Ser
305 310 315 320
Met Ile Lys Arg Tyr Asp Glu His His Gln Asp Leu Thr Leu Leu Lys
325 330 335
Ala Leu Val Arg Gln Gln Leu Pro Glu Lys Tyr Lys Glu Ile Phe Phe
340 345 350
Asp Gln Ser Lys Asn Gly Tyr Ala Gly Tyr Ile Asp Gly Gly Ala Ser
355 360 365
Gln Glu Glu Phe Tyr Lys Phe Ile Lys Pro Ile Leu Glu Lys Met Asp
370 375 380
Gly Thr Glu Glu Leu Leu Val Lys Leu Asn Arg Glu Asp Leu Leu Arg
385 390 395 400
Lys Gln Arg Thr Phe Asp Asn Gly Ser Ile Pro His Gln Ile His Leu
405 410 415
Gly Glu Leu His Ala Ile Leu Arg Arg Gln Glu Asp Phe Tyr Pro Phe
420 425 430
Leu Lys Asp Asn Arg Glu Lys Ile Glu Lys Ile Leu Thr Phe Arg Ile
435 440 445
Pro Tyr Tyr Val Gly Pro Leu Ala Arg Gly Asn Ser Arg Phe Ala Trp
450 455 460
Met Thr Arg Lys Ser Glu Glu Thr Ile Thr Pro Trp Asn Phe Glu Glu
465 470 475 480
Val Val Asp Lys Gly Ala Ser Ala Gln Ser Phe Ile Glu Arg Met Thr
485 490 495
Asn Phe Asp Lys Asn Leu Pro Asn Glu Lys Val Leu Pro Lys His Ser
500 505 510
Leu Leu Tyr Glu Tyr Phe Thr Val Tyr Asn Glu Leu Thr Lys Val Lys
515 520 525
Tyr Val Thr Glu Gly Met Arg Lys Pro Ala Phe Leu Ser Gly Glu Gln
530 535 540
Lys Lys Ala Ile Val Asp Leu Leu Phe Lys Thr Asn Arg Lys Val Thr
545 550 555 560
Val Lys Gln Leu Lys Glu Asp Tyr Phe Lys Lys Ile Glu Cys Phe Asp
565 570 575
Ser Val Glu Ile Ser Gly Val Glu Asp Arg Phe Asn Ala Ser Leu Gly
580 585 590
Thr Tyr His Asp Leu Leu Lys Ile Ile Lys Asp Lys Asp Phe Leu Asp
595 600 605
Asn Glu Glu Asn Glu Asp Ile Leu Glu Asp Ile Val Leu Thr Leu Thr
610 615 620
Leu Phe Glu Asp Arg Glu Met Ile Glu Glu Arg Leu Lys Thr Tyr Ala
625 630 635 640
His Leu Phe Asp Asp Lys Val Met Lys Gln Leu Lys Arg Arg Arg Tyr
645 650 655
Thr Gly Trp Gly Arg Leu Ser Arg Lys Leu Ile Asn Gly Ile Arg Asp
660 665 670
Lys Gln Ser Gly Lys Thr Ile Leu Asp Phe Leu Lys Ser Asp Gly Phe
675 680 685
Ala Asn Arg Asn Phe Met Gln Leu Ile His Asp Asp Ser Leu Thr Phe
690 695 700
Lys Glu Asp Ile Gln Lys Ala Gln Val Ser Gly Gln Gly Asp Ser Leu
705 710 715 720
His Glu His Ile Ala Asn Leu Ala Gly Ser Pro Ala Ile Lys Lys Gly
725 730 735
Ile Leu Gln Thr Val Lys Val Val Asp Glu Leu Val Lys Val Met Gly
740 745 750
Arg His Lys Pro Glu Asn Ile Val Ile Glu Met Ala Arg Glu Asn Gln
755 760 765
Thr Thr Gln Lys Gly Gln Lys Asn Ser Arg Glu Arg Met Lys Arg Ile
770 775 780
Glu Glu Gly Ile Lys Glu Leu Gly Ser Gln Ile Leu Lys Glu His Pro
785 790 795 800
Val Glu Asn Thr Gln Leu Gln Asn Glu Lys Leu Tyr Leu Tyr Tyr Leu
805 810 815
Gln Asn Gly Arg Asp Met Tyr Val Asp Gln Glu Leu Asp Ile Asn Arg
820 825 830
Leu Ser Asp Tyr Asp Val Ala Ala Ile Val Pro Gln Ser Phe Leu Lys
835 840 845
Asp Asp Ser Ile Asp Asn Lys Val Leu Thr Arg Ser Asp Lys Ala Arg
850 855 860
Gly Lys Ser Asp Asn Val Pro Ser Glu Glu Val Val Lys Lys Met Lys
865 870 875 880
Asn Tyr Trp Arg Gln Leu Leu Asn Ala Lys Leu Ile Thr Gln Arg Lys
885 890 895
Phe Asp Asn Leu Thr Lys Ala Glu Arg Gly Gly Leu Ser Glu Leu Asp
900 905 910
Lys Ala Gly Phe Ile Lys Arg Gln Leu Val Glu Thr Arg Gln Ile Thr
915 920 925
Lys His Val Ala Gln Ile Leu Asp Ser Arg Met Asn Thr Lys Tyr Asp
930 935 940
Glu Asn Asp Lys Leu Ile Arg Glu Val Lys Val Ile Thr Leu Lys Ser
945 950 955 960
Lys Leu Val Ser Asp Phe Arg Lys Asp Phe Gln Phe Tyr Lys Val Arg
965 970 975
Glu Ile Asn Asn Tyr His His Ala His Asp Ala Tyr Leu Asn Ala Val
980 985 990
Val Gly Thr Ala Leu Ile Lys Lys Tyr Pro Lys Leu Glu Ser Glu Phe
995 1000 1005
Val Tyr Gly Asp Tyr Lys Val Tyr Asp Val Arg Lys Met Ile Ala
1010 1015 1020
Lys Ser Glu Gln Glu Ile Gly Lys Ala Thr Ala Lys Tyr Phe Phe
1025 1030 1035
Tyr Ser Asn Ile Met Asn Phe Phe Lys Thr Glu Ile Thr Leu Ala
1040 1045 1050
Asn Gly Glu Ile Arg Lys Arg Pro Leu Ile Glu Thr Asn Gly Glu
1055 1060 1065
Thr Gly Glu Ile Val Trp Asp Lys Gly Arg Asp Phe Ala Thr Val
1070 1075 1080
Arg Lys Val Leu Ser Met Pro Gln Val Asn Ile Val Lys Lys Thr
1085 1090 1095
Glu Val Gln Thr Gly Gly Phe Ser Lys Glu Ser Ile Leu Pro Lys
1100 1105 1110
Arg Asn Ser Asp Lys Leu Ile Ala Arg Lys Lys Asp Trp Asp Pro
1115 1120 1125
Lys Lys Tyr Gly Gly Phe Asp Ser Pro Thr Val Ala Tyr Ser Val
1130 1135 1140
Leu Val Val Ala Lys Val Glu Lys Gly Lys Ser Lys Lys Leu Lys
1145 1150 1155
Ser Val Lys Glu Leu Leu Gly Ile Thr Ile Met Glu Arg Ser Ser
1160 1165 1170
Phe Glu Lys Asn Pro Ile Asp Phe Leu Glu Ala Lys Gly Tyr Lys
1175 1180 1185
Glu Val Lys Lys Asp Leu Ile Ile Lys Leu Pro Lys Tyr Ser Leu
1190 1195 1200
Phe Glu Leu Glu Asn Gly Arg Lys Arg Met Leu Ala Ser Ala Gly
1205 1210 1215
Glu Leu Gln Lys Gly Asn Glu Leu Ala Leu Pro Ser Lys Tyr Val
1220 1225 1230
Asn Phe Leu Tyr Leu Ala Ser His Tyr Glu Lys Leu Lys Gly Ser
1235 1240 1245
Pro Glu Asp Asn Glu Gln Lys Gln Leu Phe Val Glu Gln His Lys
1250 1255 1260
His Tyr Leu Asp Glu Ile Ile Glu Gln Ile Ser Glu Phe Ser Lys
1265 1270 1275
Arg Val Ile Leu Ala Asp Ala Asn Leu Asp Lys Val Leu Ser Ala
1280 1285 1290
Tyr Asn Lys His Arg Asp Lys Pro Ile Arg Glu Gln Ala Glu Asn
1295 1300 1305
Ile Ile His Leu Phe Thr Leu Thr Asn Leu Gly Ala Pro Ala Ala
1310 1315 1320
Phe Lys Tyr Phe Asp Thr Thr Ile Asp Arg Lys Arg Tyr Thr Ser
1325 1330 1335
Thr Lys Glu Val Leu Asp Ala Thr Leu Ile His Gln Ser Ile Thr
1340 1345 1350
Gly Leu Tyr Glu Thr Arg Ile Asp Leu Ser Gln Leu Gly Gly Asp
1355 1360 1365
<210> 72
<211> 594
<212> PRT
<213> Artificial Sequence
<220>
<223> hyperactive PiggyBac (PB) transposase amino acid
<400> 72
Met Gly Ser Ser Leu Asp Asp Glu His Ile Leu Ser Ala Leu Leu Gln
1 5 10 15
Ser Asp Asp Glu Leu Val Gly Glu Asp Ser Asp Ser Glu Val Ser Asp
20 25 30
His Val Ser Glu Asp Asp Val Gln Ser Asp Thr Glu Glu Ala Phe Ile
35 40 45
Asp Glu Val His Glu Val Gln Pro Thr Ser Ser Gly Ser Glu Ile Leu
50 55 60
Asp Glu Gln Asn Val Ile Glu Gln Pro Gly Ser Ser Leu Ala Ser Asn
65 70 75 80
Arg Ile Leu Thr Leu Pro Gln Arg Thr Ile Arg Gly Lys Asn Lys His
85 90 95
Cys Trp Ser Thr Ser Lys Pro Thr Arg Arg Ser Arg Val Ser Ala Leu
100 105 110
Asn Ile Val Arg Ser Gln Arg Gly Pro Thr Arg Met Cys Arg Asn Ile
115 120 125
Tyr Asp Pro Leu Leu Cys Phe Lys Leu Phe Phe Thr Asp Glu Ile Ile
130 135 140
Ser Glu Ile Val Lys Trp Thr Asn Ala Glu Ile Ser Leu Lys Arg Arg
145 150 155 160
Glu Ser Met Thr Ser Ala Thr Phe Arg Asp Thr Asn Glu Asp Glu Ile
165 170 175
Tyr Ala Phe Phe Gly Ile Leu Val Met Thr Ala Val Arg Lys Asp Asn
180 185 190
His Met Ser Thr Asp Asp Leu Phe Asp Arg Ser Leu Ser Met Val Tyr
195 200 205
Val Ser Val Met Ser Arg Asp Arg Phe Asp Phe Leu Ile Arg Cys Leu
210 215 220
Arg Met Asp Asp Lys Ser Ile Arg Pro Thr Leu Arg Glu Asn Asp Val
225 230 235 240
Phe Thr Pro Val Arg Lys Ile Trp Asp Leu Phe Ile His Gln Cys Ile
245 250 255
Gln Asn Tyr Thr Pro Gly Ala His Leu Thr Ile Asp Glu Gln Leu Leu
260 265 270
Gly Phe Arg Gly Arg Cys Pro Phe Arg Val Tyr Ile Pro Asn Lys Pro
275 280 285
Ser Lys Tyr Gly Ile Lys Ile Leu Met Met Cys Asp Ser Gly Thr Lys
290 295 300
Tyr Met Ile Asn Gly Met Pro Tyr Leu Gly Arg Gly Thr Gln Thr Asn
305 310 315 320
Gly Val Pro Leu Gly Glu Tyr Tyr Val Lys Glu Leu Ser Lys Pro Val
325 330 335
His Gly Ser Cys Arg Asn Ile Thr Cys Asp Asn Trp Phe Thr Ser Ile
340 345 350
Pro Leu Ala Lys Asn Leu Leu Gln Glu Pro Tyr Lys Leu Thr Ile Val
355 360 365
Gly Thr Val Arg Ser Asn Lys Arg Glu Ile Pro Glu Val Leu Lys Asn
370 375 380
Ser Arg Ser Arg Pro Val Gly Thr Ser Met Phe Cys Phe Asp Gly Pro
385 390 395 400
Leu Thr Leu Val Ser Tyr Lys Pro Lys Pro Ala Lys Met Val Tyr Leu
405 410 415
Leu Ser Ser Cys Asp Glu Asp Ala Ser Ile Asn Glu Ser Thr Gly Lys
420 425 430
Pro Gln Met Val Met Tyr Tyr Asn Gln Thr Lys Gly Gly Val Asp Thr
435 440 445
Leu Asp Gln Met Cys Ser Val Met Thr Cys Ser Arg Lys Thr Asn Arg
450 455 460
Trp Pro Met Ala Leu Leu Tyr Gly Met Ile Asn Ile Ala Cys Ile Asn
465 470 475 480
Ser Phe Ile Ile Tyr Ser His Asn Val Ser Ser Lys Gly Glu Lys Val
485 490 495
Gln Ser Arg Lys Lys Phe Met Arg Asn Leu Tyr Met Gly Leu Thr Ser
500 505 510
Ser Phe Met Arg Lys Arg Leu Glu Ala Pro Thr Leu Lys Arg Tyr Leu
515 520 525
Arg Asp Asn Ile Ser Asn Ile Leu Pro Lys Glu Val Pro Gly Thr Ser
530 535 540
Asp Asp Ser Thr Glu Glu Pro Val Met Lys Lys Arg Thr Tyr Cys Thr
545 550 555 560
Tyr Cys Pro Ser Lys Ile Arg Arg Lys Ala Ser Ala Ser Cys Lys Lys
565 570 575
Cys Lys Lys Val Ile Cys Arg Glu His Asn Ile Asp Met Cys Gln Ser
580 585 590
Cys Phe
<210> 73
<211> 340
<212> PRT
<213> Artificial Sequence
<220>
<223> hyperactive Sleeping Beauty (SB100) transposase amino acid
<400> 73
Met Gly Lys Ser Lys Glu Ile Ser Gln Asp Leu Arg Lys Arg Ile Val
1 5 10 15
Asp Leu His Lys Ser Gly Ser Ser Leu Gly Ala Ile Ser Lys Arg Leu
20 25 30
Ala Val Pro Arg Ser Ser Val Gln Thr Ile Val Arg Lys Tyr Lys His
35 40 45
His Gly Thr Thr Gln Pro Ser Tyr Arg Ser Gly Arg Arg Arg Val Leu
50 55 60
Ser Pro Arg Asp Glu Arg Thr Leu Val Arg Lys Val Gln Ile Asn Pro
65 70 75 80
Arg Thr Thr Ala Lys Asp Leu Val Lys Met Leu Glu Glu Thr Gly Thr
85 90 95
Lys Val Ser Ile Ser Thr Val Lys Arg Val Leu Tyr Arg His Asn Leu
100 105 110
Lys Gly His Ser Ala Arg Lys Lys Pro Leu Leu Gln Asn Arg His Lys
115 120 125
Lys Ala Arg Leu Arg Phe Ala Thr Ala His Gly Asp Lys Asp Arg Thr
130 135 140
Phe Trp Arg Asn Val Leu Trp Ser Asp Glu Thr Lys Ile Glu Leu Phe
145 150 155 160
Gly His Asn Asp His Arg Tyr Val Trp Arg Lys Lys Gly Glu Ala Cys
165 170 175
Lys Pro Lys Asn Thr Ile Pro Thr Val Lys His Gly Gly Gly Ser Ile
180 185 190
Met Leu Trp Gly Cys Phe Ala Ala Gly Gly Thr Gly Ala Leu His Lys
195 200 205
Ile Asp Gly Ile Met Asp Ala Val Gln Tyr Val Asp Ile Leu Lys Gln
210 215 220
His Leu Lys Thr Ser Val Arg Lys Leu Lys Leu Gly Arg Lys Trp Val
225 230 235 240
Phe Gln His Asp Asn Asp Pro Lys His Thr Ser Lys Val Val Ala Lys
245 250 255
Trp Leu Lys Asp Asn Lys Val Lys Val Leu Glu Trp Pro Ser Gln Ser
260 265 270
Pro Asp Leu Asn Pro Ile Glu Asn Leu Trp Ala Glu Leu Lys Lys Arg
275 280 285
Val Arg Ala Arg Arg Pro Thr Asn Leu Thr Gln Leu His Gln Leu Cys
290 295 300
Gln Glu Glu Trp Ala Lys Ile His Pro Asn Tyr Cys Gly Lys Leu Val
305 310 315 320
Glu Gly Tyr Pro Lys Arg Leu Thr Gln Val Lys Gln Phe Lys Gly Asn
325 330 335
Ala Thr Lys Tyr
340
<210> 74
<211> 118
<212> DNA
<213> Artificial Sequence
<220>
<223> IN cPPT/CTS domain Nucleic Acid
<400> 74
ttttaaaaga aaagggggga ttggggggta cagtgcaggg gaaagaatag tagacataat 60
agcaacagac atacaaacta aagaattaca aaaacaaatt acaaaaattc aaaatttt 118
<210> 75
<211> 39
<212> PRT
<213> Artificial Sequence
<220>
<223> Primer GG-cPPT-Fw
<400> 75
Thr Cys Cys Thr Cys Thr Cys Gly Thr Cys Thr Cys Cys Ala Thr Thr
1 5 10 15
Ala Thr Thr Thr Thr Ala Ala Ala Ala Gly Ala Ala Ala Ala Gly Gly
20 25 30
Gly Gly Gly Gly Ala Thr Thr
35
<210> 76
<211> 39
<212> PRT
<213> Artificial Sequence
<220>
<223> Primer GG-cPPT-STOP-Fw
<400> 76
Thr Cys Cys Thr Cys Thr Cys Gly Thr Cys Thr Cys Cys Ala Thr Thr
1 5 10 15
Ala Ala Thr Thr Thr Ala Ala Ala Ala Gly Ala Ala Ala Ala Gly Gly
20 25 30
Gly Gly Gly Gly Ala Thr Thr
35
<210> 77
<211> 47
<212> PRT
<213> Artificial Sequence
<220>
<223> Primer GG-cPPT-Rv
<400> 77
Thr Cys Cys Thr Cys Thr Cys Gly Thr Cys Thr Cys Cys Cys Thr Gly
1 5 10 15
Ala Ala Ala Ala Ala Thr Thr Thr Thr Gly Ala Ala Thr Thr Thr Thr
20 25 30
Thr Gly Thr Ala Ala Thr Thr Thr Gly Thr Thr Thr Thr Thr Gly
35 40 45
<210> 78
<211> 39
<212> PRT
<213> Artificial Sequence
<220>
<223> Primer GG-AAVS1-6d-Fw
<400> 78
Thr Cys Cys Thr Cys Thr Cys Gly Thr Cys Thr Cys Cys Ala Thr Thr
1 5 10 15
Ala Thr Ala Thr Gly Gly Cys Thr Cys Cys Ala Ala Ala Gly Ala Ala
20 25 30
Ala Ala Ala Gly Ala Gly Gly
35
<210> 79
<211> 45
<212> PRT
<213> Artificial Sequence
<220>
<223> Primer GG-AAVS1-6d-Rv
<400> 79
Thr Cys Cys Thr Cys Thr Cys Gly Thr Cys Thr Cys Cys Cys Thr Gly
1 5 10 15
Ala Thr Cys Ala Ala Thr Cys Cys Thr Cys Ala Thr Cys Cys Thr Gly
20 25 30
Thr Cys Thr Ala Cys Thr Thr Gly Cys Cys Ala Cys Ala
35 40 45
<210> 80
<211> 35
<212> PRT
<213> Artificial Sequence
<220>
<223> Primer GG-AAVS1-6d (-NLS)-Fw
<400> 80
Thr Cys Cys Thr Cys Thr Cys Gly Thr Cys Thr Cys Cys Ala Thr Thr
1 5 10 15
Ala Thr Ala Thr Gly Gly Cys Cys Cys Ala Gly Gly Cys Thr Gly Cys
20 25 30
Thr Cys Thr
35
<210> 81
<211> 24
<212> PRT
<213> Artificial Sequence
<220>
<223> Primer IN-Fw
<400> 81
Thr Thr Thr Thr Ala Gly Ala Thr Gly Gly Ala Ala Thr Ala Gly Ala
1 5 10 15
Thr Ala Ala Gly Gly Cys Cys Cys
20
<210> 82
<211> 32
<212> PRT
<213> Artificial Sequence
<220>
<223> Primer XbaI-pSICO_IC-5'Fw1
<400> 82
Cys Thr Ala Gly Cys Thr Cys Thr Ala Gly Ala Thr Gly Gly Cys Thr
1 5 10 15
Ala Ala Cys Thr Ala Gly Gly Gly Ala Ala Cys Cys Cys Ala Cys Thr
20 25 30
<210> 83
<211> 33
<212> PRT
<213> Artificial Sequence
<220>
<223> Primer SacI-pSICO_IC-5'Rv1
<400> 83
Cys Thr Ala Gly Cys Gly Ala Gly Cys Thr Cys Cys Cys Ala Gly Gly
1 5 10 15
Cys Thr Cys Ala Gly Ala Thr Cys Thr Gly Gly Thr Cys Thr Ala Ala
20 25 30
Cys
<210> 84
<211> 31
<212> PRT
<213> Artificial Sequence
<220>
<223> Primer XbaI-pSICO_IC-5'Fw2
<400> 84
Cys Thr Ala Gly Cys Thr Cys Thr Ala Gly Ala Cys Thr Ala Ala Cys
1 5 10 15
Thr Ala Gly Gly Gly Ala Ala Cys Cys Cys Ala Cys Thr Gly Cys
20 25 30
<210> 85
<211> 48
<212> PRT
<213> Artificial Sequence
<220>
<223> Primer SacI-pSICO_IC-5'Rv2
<400> 85
Cys Cys Thr Cys Thr Cys Thr Ala Thr Gly Gly Gly Cys Ala Gly Thr
1 5 10 15
Cys Thr Ala Gly Cys Gly Ala Gly Cys Thr Cys Cys Thr Gly Gly Thr
20 25 30
Cys Thr Ala Ala Cys Cys Ala Gly Ala Gly Ala Gly Ala Cys Cys Cys
35 40 45
<210> 86
<211> 32
<212> PRT
<213> Artificial Sequence
<220>
<223> Primer XbaI-pSICO_IC-3'Fw1
<400> 86
Cys Thr Ala Gly Cys Thr Cys Thr Ala Gly Ala Thr Cys Cys Cys Thr
1 5 10 15
Cys Ala Gly Ala Cys Cys Cys Thr Thr Thr Thr Ala Gly Thr Cys Ala
20 25 30
<210> 87
<211> 31
<212> PRT
<213> Artificial Sequence
<220>
<223> Primer SacI-pSICO_IC-3'Rv1
<400> 87
Cys Thr Ala Gly Cys Gly Ala Gly Cys Thr Cys Cys Ala Ala Cys Ala
1 5 10 15
Gly Ala Cys Gly Gly Gly Cys Ala Cys Ala Cys Ala Cys Thr Ala
20 25 30
<210> 88
<211> 32
<212> PRT
<213> Artificial Sequence
<220>
<223> Primer XbaI-pSICO_IC-3'Fw2
<400> 88
Cys Thr Ala Gly Cys Thr Cys Thr Ala Gly Ala Ala Ala Ala Ala Thr
1 5 10 15
Cys Thr Cys Thr Ala Gly Cys Ala Gly Cys Cys Cys Ala Thr Cys Cys
20 25 30
<210> 89
<211> 47
<212> PRT
<213> Artificial Sequence
<220>
<223> Primer SacI-pSICO_IC-3'Rv2
<400> 89
Cys Cys Thr Cys Thr Cys Thr Ala Thr Gly Gly Gly Cys Ala Gly Thr
1 5 10 15
Cys Thr Ala Gly Cys Gly Ala Gly Cys Thr Cys Gly Ala Cys Gly Gly
20 25 30
Gly Cys Ala Cys Ala Cys Ala Cys Thr Ala Cys Thr Thr Gly Ala
35 40 45
<210> 90
<211> 35
<212> PRT
<213> Artificial Sequence
<220>
<223> Primer CCD1-A128T-F
<400> 90
Thr Cys Ala Cys Cys Ala Gly Thr Ala Cys Thr Ala Cys Ala Gly Thr
1 5 10 15
Thr Ala Ala Gly Ala Cys Cys Gly Cys Cys Thr Gly Thr Thr Gly Gly
20 25 30
Thr Gly Gly
35
<210> 91
<211> 35
<212> PRT
<213> Artificial Sequence
<220>
<223> Primer CCD1-A128T-R
<400> 91
Cys Cys Ala Cys Cys Ala Ala Cys Ala Gly Gly Cys Gly Gly Thr Cys
1 5 10 15
Thr Thr Ala Ala Cys Thr Gly Thr Ala Gly Thr Ala Cys Thr Gly Gly
20 25 30
Thr Gly Ala
35
<210> 92
<211> 41
<212> PRT
<213> Artificial Sequence
<220>
<223> Primer CCD2-E170G-F
<400> 92
Ala Cys Ala Gly Gly Thr Ala Ala Gly Ala Gly Ala Thr Cys Ala Gly
1 5 10 15
Gly Cys Thr Gly Gly Cys Cys Ala Thr Cys Thr Thr Ala Ala Gly Ala
20 25 30
Cys Ala Gly Cys Ala Gly Thr Ala Cys
35 40
<210> 93
<211> 41
<212> PRT
<213> Artificial Sequence
<220>
<223> Primer CCD2-E170G-R
<400> 93
Gly Thr Ala Cys Thr Gly Cys Thr Gly Thr Cys Thr Thr Ala Ala Gly
1 5 10 15
Ala Thr Gly Gly Cys Cys Ala Gly Cys Cys Thr Gly Ala Thr Cys Thr
20 25 30
Cys Thr Thr Ala Cys Cys Thr Gly Thr
35 40
<210> 94
<211> 63
<212> PRT
<213> Artificial Sequence
<220>
<223> Primer NTD1-E10/13K-F
<400> 94
Gly Gly Thr Thr Thr Thr Thr Thr Ala Gly Ala Thr Gly Gly Ala Ala
1 5 10 15
Thr Ala Gly Ala Thr Ala Ala Gly Gly Cys Cys Cys Ala Ala Ala Ala
20 25 30
Gly Gly Ala Ala Cys Ala Thr Ala Ala Gly Ala Ala Ala Thr Ala Thr
35 40 45
Cys Ala Cys Ala Gly Thr Ala Ala Thr Thr Gly Gly Ala Gly Ala
50 55 60
<210> 95
<211> 63
<212> PRT
<213> Artificial Sequence
<220>
<223> Primer NTD1-E10/13K-R
<400> 95
Thr Cys Thr Cys Cys Ala Ala Thr Thr Ala Cys Thr Gly Thr Gly Ala
1 5 10 15
Thr Ala Thr Thr Thr Cys Thr Thr Ala Thr Gly Thr Thr Cys Cys Thr
20 25 30
Thr Thr Thr Gly Gly Gly Cys Cys Thr Thr Ala Thr Cys Thr Ala Thr
35 40 45
Thr Cys Cys Ala Thr Cys Thr Ala Ala Ala Ala Ala Ala Cys Cys
50 55 60
<210> 96
<211> 49
<212> PRT
<213> Artificial Sequence
<220>
<223> Primer Solubility-F185K-F
<400> 96
Ala Ala Ala Thr Gly Gly Cys Ala Gly Thr Ala Thr Thr Cys Ala Thr
1 5 10 15
Cys Cys Ala Cys Ala Ala Thr Ala Ala Gly Ala Ala Ala Ala Gly Ala
20 25 30
Ala Ala Ala Gly Gly Gly Gly Gly Gly Ala Thr Thr Gly Gly Gly Gly
35 40 45
Gly
<210> 97
<211> 49
<212> PRT
<213> Artificial Sequence
<220>
<223> Primer Solubility-F185K-R
<400> 97
Cys Cys Cys Cys Cys Ala Ala Thr Cys Cys Cys Cys Cys Cys Thr Thr
1 5 10 15
Thr Thr Cys Thr Thr Thr Thr Cys Thr Thr Ala Thr Thr Gly Thr Gly
20 25 30
Gly Ala Thr Gly Ala Ala Thr Ala Cys Thr Gly Cys Cys Ala Thr Thr
35 40 45
Thr
<210> 98
<211> 54
<212> PRT
<213> Artificial Sequence
<220>
<223> Primer Primer NGS-aavs fw
<400> 98
Ala Cys Ala Cys Thr Cys Thr Thr Thr Cys Cys Cys Thr Ala Cys Ala
1 5 10 15
Cys Gly Ala Cys Gly Cys Thr Cys Thr Thr Cys Cys Gly Ala Thr Cys
20 25 30
Thr Ala Gly Gly Ala Cys Ala Gly Cys Ala Thr Gly Thr Thr Thr Gly
35 40 45
Cys Thr Gly Cys Cys Thr
50
<210> 99
<211> 51
<212> PRT
<213> Artificial Sequence
<220>
<223> Primer NGS-aavs rv
<400> 99
Gly Ala Cys Thr Gly Gly Ala Gly Thr Thr Cys Ala Gly Ala Cys Gly
1 5 10 15
Thr Gly Thr Gly Cys Thr Cys Thr Thr Cys Cys Gly Ala Thr Cys Thr
20 25 30
Gly Cys Thr Cys Cys Ala Gly Gly Ala Ala Ala Thr Gly Gly Gly Gly
35 40 45
Gly Thr Gly
50
<210> 100
<211> 34
<212> PRT
<213> Artificial Sequence
<220>
<223> Primer PB R245A
<400> 100
Cys Gly Thr Gly Thr Thr Cys Ala Cys Cys Cys Cys Cys Gly Thr Gly
1 5 10 15
Gly Cys Ala Ala Ala Gly Ala Thr Cys Thr Gly Gly Gly Ala Cys Cys
20 25 30
Thr Gly
<210> 101
<211> 36
<212> PRT
<213> Artificial Sequence
<220>
<223> Primer PB R275-277A
<400> 101
Ala Gly Cys Thr Gly Cys Thr Gly Gly Gly Cys Thr Thr Cys Gly Cys
1 5 10 15
Gly Gly Gly Cys Gly Cys Gly Thr Gly Cys Cys Cys Cys Thr Thr Cys
20 25 30
Ala Gly Gly Gly
35
<210> 102
<211> 28
<212> PRT
<213> Artificial Sequence
<220>
<223> Primer PB R388A
<400> 102
Gly Ala Ala Cys Ala Gly Cys Ala Gly Gly Thr Cys Cys Gly Cys Gly
1 5 10 15
Cys Cys Cys Gly Thr Gly Gly Gly Cys Ala Cys Cys
20 25
<210> 103
<211> 32
<212> PRT
<213> Artificial Sequence
<220>
<223> Primer PB S351A
<400> 103
Gly Ala Cys Ala Ala Cys Thr Gly Gly Thr Thr Cys Ala Cys Cys Gly
1 5 10 15
Cys Cys Ala Thr Cys Cys Cys Cys Cys Thr Gly Gly Cys Cys Ala Ala
20 25 30
<210> 104
<211> 30
<212> PRT
<213> Artificial Sequence
<220>
<223> Primer PB W465A
<400> 104
Gly Ala Ala Ala Gly Ala Cys Cys Ala Ala Cys Ala Gly Gly Gly Cys
1 5 10 15
Gly Cys Cys Cys Ala Thr Gly Gly Cys Cys Cys Thr Gly Cys
20 25 30
<210> 105
<211> 43
<212> PRT
<213> Artificial Sequence
<220>
<223> Primer PB R372A-K375A
<400> 105
Cys Ala Thr Cys Gly Thr Gly Gly Gly Cys Ala Cys Cys Gly Thr Gly
1 5 10 15
Gly Cys Ala Ala Gly Cys Ala Ala Cys Gly Cys Gly Ala Gly Ala Gly
20 25 30
Ala Gly Ala Thr Cys Cys Cys Cys Gly Ala Gly
35 40
<210> 106
<211> 29
<212> PRT
<213> Artificial Sequence
<220>
<223> Primer PB D450N
<400> 106
Gly Cys Gly Thr Gly Gly Ala Cys Ala Cys Cys Cys Thr Gly Ala Ala
1 5 10 15
Cys Cys Ala Gly Ala Thr Gly Thr Gly Cys Ala Gly Cys
20 25
<210> 107
<211> 21
<212> PRT
<213> Artificial Sequence
<220>
<223> Primer SYBR-WPRE-3_Fw
<400> 107
Ala Cys Gly Cys Thr Ala Thr Gly Thr Gly Gly Ala Thr Ala Cys Gly
1 5 10 15
Cys Thr Gly Cys Thr
20
<210> 108
<211> 21
<212> PRT
<213> Artificial Sequence
<220>
<223> Primer SYBR-WPRE-3_Rv
<400> 108
Ala Gly Cys Ala Ala Ala Cys Ala Cys Ala Gly Thr Gly Cys Ala Cys
1 5 10 15
Ala Cys Cys Ala Cys
20
<210> 109
<211> 20
<212> PRT
<213> Artificial Sequence
<220>
<223> Primer SYBR-RNaseP_Fw
<400> 109
Gly Gly Ala Gly Thr Gly Ala Gly Gly Ala Gly Gly Gly Ala Thr Gly
1 5 10 15
Thr Gly Ala Ala
20
<210> 110
<211> 20
<212> PRT
<213> Artificial Sequence
<220>
<223> Primer SYBR-RNaseP_Rv
<400> 110
Ala Thr Thr Gly Ala Gly Gly Gly Cys Ala Cys Thr Gly Gly Ala Ala
1 5 10 15
Ala Thr Thr Gly
20
<210> 111
<211> 68
<212> PRT
<213> Artificial Sequence
<220>
<223> Primer Illumina custom
<400> 111
Ala Ala Thr Gly Ala Thr Ala Cys Gly Gly Cys Gly Ala Cys Cys Ala
1 5 10 15
Cys Cys Gly Ala Gly Ala Thr Cys Thr Ala Cys Ala Cys Ala Gly Cys
20 25 30
Thr Ala Gly Ala Cys Ala Cys Thr Cys Thr Thr Thr Cys Cys Cys Thr
35 40 45
Ala Cys Ala Cys Gly Ala Cys Gly Cys Thr Cys Thr Thr Cys Cys Gly
50 55 60
Ala Thr Cys Thr
65
<210> 112
<211> 64
<212> PRT
<213> Artificial Sequence
<220>
<223> Primer NEBNext Index 9
<400> 112
Cys Ala Ala Gly Cys Ala Gly Ala Ala Gly Ala Cys Gly Gly Cys Ala
1 5 10 15
Thr Ala Cys Gly Ala Gly Ala Thr Cys Thr Gly Ala Thr Cys Gly Thr
20 25 30
Gly Ala Cys Thr Gly Gly Ala Gly Thr Thr Cys Ala Gly Ala Cys Gly
35 40 45
Thr Gly Thr Gly Cys Thr Cys Thr Thr Cys Cys Gly Ala Thr Cys Thr
50 55 60
<210> 113
<211> 53
<212> PRT
<213> Artificial Sequence
<220>
<223> Primer NGS cluster 1 fw
<400> 113
Ala Cys Ala Cys Thr Cys Thr Thr Thr Cys Cys Cys Thr Ala Cys Ala
1 5 10 15
Cys Gly Ala Cys Gly Cys Thr Cys Thr Thr Cys Cys Gly Ala Thr Cys
20 25 30
Thr Cys Thr Gly Cys Gly Gly Gly Ala Gly Ala Ala Cys Gly Ala Cys
35 40 45
Gly Thr Gly Thr Thr
50
<210> 114
<211> 55
<212> PRT
<213> Artificial Sequence
<220>
<223> Primer NGS cluster 2 rv
<400> 114
Gly Ala Cys Thr Gly Gly Ala Gly Thr Thr Cys Ala Gly Ala Cys Gly
1 5 10 15
Thr Gly Thr Gly Cys Thr Cys Thr Thr Cys Cys Gly Ala Thr Cys Thr
20 25 30
Cys Cys Thr Cys Ala Cys Cys Thr Thr Cys Cys Thr Cys Thr Thr Cys
35 40 45
Thr Thr Cys Thr Thr Gly Gly
50 55
<210> 115
<211> 42
<212> PRT
<213> Artificial Sequence
<220>
<223> Primer CMV-F
<400> 115
Cys Thr Gly Cys Ala Gly Cys Gly Cys Gly Gly Gly Gly Ala Thr Cys
1 5 10 15
Thr Cys Ala Thr Gly Cys Thr Gly Gly Ala Gly Thr Thr Cys Thr Thr
20 25 30
Cys Gly Cys Cys Cys Ala Cys Cys Cys Cys
35 40
<210> 116
<211> 25
<212> PRT
<213> Artificial Sequence
<220>
<223> Primer cas9 rv
<400> 116
Cys Ala Cys Cys Thr Thr Cys Cys Thr Cys Thr Thr Cys Thr Thr Cys
1 5 10 15
Thr Thr Gly Gly Gly Gly Thr Cys Ala
20 25
<210> 117
<211> 573
<212> PRT
<213> Artificial Sequence
<220>
<223> ZFP_TCRa4 na sequence
<400> 117
Ala Thr Gly Gly Cys Thr Cys Cys Thr Ala Ala Gly Ala Ala Gly Ala
1 5 10 15
Ala Gly Cys Gly Gly Ala Ala Ala Gly Thr Cys Gly Gly Cys Ala Thr
20 25 30
Ala Cys Ala Cys Gly Gly Ala Gly Thr Gly Cys Cys Thr Gly Cys Thr
35 40 45
Gly Cys Ala Ala Thr Gly Gly Cys Ala Gly Ala Ala Ala Gly Gly Cys
50 55 60
Cys Ala Thr Thr Cys Cys Ala Ala Thr Gly Cys Ala Gly Ala Ala Thr
65 70 75 80
Ala Thr Gly Cys Ala Thr Gly Ala Gly Gly Ala Ala Cys Thr Thr Cys
85 90 95
Thr Cys Ala Gly Ala Thr Cys Gly Cys Ala Gly Thr Ala Ala Cys Cys
100 105 110
Thr Cys Thr Cys Ala Ala Gly Gly Cys Ala Thr Ala Thr Ala Cys Gly
115 120 125
Gly Ala Cys Cys Cys Ala Thr Ala Cys Gly Gly Gly Gly Gly Ala Ala
130 135 140
Ala Ala Ala Cys Cys Ala Thr Thr Thr Gly Cys Cys Thr Gly Thr Gly
145 150 155 160
Ala Thr Ala Thr Ala Thr Gly Thr Gly Gly Cys Cys Gly Cys Ala Ala
165 170 175
Gly Thr Thr Cys Gly Cys Thr Cys Ala Gly Ala Ala Ala Gly Thr Gly
180 185 190
Ala Cys Cys Thr Thr Gly Gly Cys Ala Gly Cys Thr Cys Ala Cys Ala
195 200 205
Cys Thr Ala Ala Gly Ala Thr Thr Cys Ala Cys Ala Cys Ala Cys Ala
210 215 220
Thr Cys Cys Ala Ala Gly Ala Gly Cys Cys Cys Cys Thr Ala Thr Cys
225 230 235 240
Cys Cys Thr Ala Ala Gly Cys Cys Gly Thr Thr Cys Cys Ala Ala Thr
245 250 255
Gly Thr Ala Gly Gly Ala Thr Ala Thr Gly Cys Ala Thr Gly Cys Gly
260 265 270
Ala Ala Ala Cys Thr Thr Cys Thr Cys Thr Gly Ala Thr Cys Gly Gly
275 280 285
Ala Gly Thr Gly Cys Ala Cys Thr Gly Ala Gly Thr Ala Gly Gly Cys
290 295 300
Ala Cys Ala Thr Cys Ala Gly Ala Ala Cys Ala Cys Ala Cys Ala Cys
305 310 315 320
Gly Gly Gly Ala Gly Ala Ala Ala Ala Gly Cys Cys Thr Thr Thr Cys
325 330 335
Gly Cys Thr Thr Gly Cys Gly Ala Thr Ala Thr Cys Thr Gly Cys Gly
340 345 350
Gly Gly Cys Gly Gly Ala Ala Gly Thr Thr Cys Gly Cys Ala Ala Cys
355 360 365
Ala Thr Cys Cys Gly Gly Gly Ala Ala Thr Cys Thr Cys Ala Cys Thr
370 375 380
Cys Gly Cys Cys Ala Thr Ala Cys Gly Ala Ala Ala Ala Thr Ala Cys
385 390 395 400
Ala Cys Ala Cys Thr Gly Gly Cys Ala Gly Cys Cys Ala Ala Ala Ala
405 410 415
Ala Cys Cys Thr Thr Thr Cys Cys Ala Ala Thr Gly Cys Cys Gly Ala
420 425 430
Ala Thr Ala Thr Gly Thr Ala Thr Gly Ala Gly Ala Ala Ala Thr Thr
435 440 445
Thr Thr Ala Gly Cys Thr Ala Cys Ala Gly Ala Ala Gly Thr Thr Cys
450 455 460
Ala Thr Thr Gly Ala Ala Ala Gly Ala Ala Cys Ala Cys Ala Thr Thr
465 470 475 480
Ala Gly Ala Ala Cys Cys Cys Ala Thr Ala Cys Cys Gly Gly Ala Gly
485 490 495
Ala Ala Ala Ala Gly Cys Cys Gly Thr Thr Cys Gly Cys Gly Thr Gly
500 505 510
Cys Gly Ala Thr Ala Thr Cys Thr Gly Cys Gly Gly Thr Cys Gly Gly
515 520 525
Ala Ala Gly Thr Thr Cys Gly Cys Thr Ala Cys Cys Thr Cys Ala Gly
530 535 540
Gly Cys Ala Ala Cys Cys Thr Gly Ala Cys Ala Cys Gly Cys Cys Ala
545 550 555 560
Cys Ala Cys Gly Ala Ala Ala Ala Thr Cys Cys Ala Cys
565 570
<210> 118
<211> 191
<212> PRT
<213> Artificial Sequence
<220>
<223> ZFP_TCRa4 aa sequence
<400> 118
Met Ala Pro Lys Lys Lys Arg Lys Val Gly Ile His Gly Val Pro Ala
1 5 10 15
Ala Met Ala Glu Arg Pro Phe Gln Cys Arg Ile Cys Met Arg Asn Phe
20 25 30
Ser Asp Arg Ser Asn Leu Ser Arg His Ile Arg Thr His Thr Gly Glu
35 40 45
Lys Pro Phe Ala Cys Asp Ile Cys Gly Arg Lys Phe Ala Gln Lys Val
50 55 60
Thr Leu Ala Ala His Thr Lys Ile His Thr His Pro Arg Ala Pro Ile
65 70 75 80
Pro Lys Pro Phe Gln Cys Arg Ile Cys Met Arg Asn Phe Ser Asp Arg
85 90 95
Ser Ala Leu Ser Arg His Ile Arg Thr His Thr Gly Glu Lys Pro Phe
100 105 110
Ala Cys Asp Ile Cys Gly Arg Lys Phe Ala Thr Ser Gly Asn Leu Thr
115 120 125
Arg His Thr Lys Ile His Thr Gly Ser Gln Lys Pro Phe Gln Cys Arg
130 135 140
Ile Cys Met Arg Asn Phe Ser Tyr Arg Ser Ser Leu Lys Glu His Ile
145 150 155 160
Arg Thr His Thr Gly Glu Lys Pro Phe Ala Cys Asp Ile Cys Gly Arg
165 170 175
Lys Phe Ala Thr Ser Gly Asn Leu Thr Arg His Thr Lys Ile His
180 185 190
<210> 119
<211> 594
<212> PRT
<213> Artificial Sequence
<220>
<223> Modified hyperactive PiggyBac aa sequence
<220>
<221> MISC_FEATURE
<222> (245)..(245)
<223> Wherein amino acid could be Ala
<220>
<221> MISC_FEATURE
<222> (275)..(275)
<223> Wherein amino acid could be Ala
<220>
<221> MISC_FEATURE
<222> (277)..(277)
<223> Wherein amino acid could be Ala
<220>
<221> MISC_FEATURE
<222> (325)..(325)
<223> Wherein amino acid could be Ala
<220>
<221> MISC_FEATURE
<222> (347)..(347)
<223> Wherein amino acid could be Ala, Ser
<220>
<221> MISC_FEATURE
<222> (351)..(351)
<223> Wherein amino acid could be Glu, Pro, Ala
<220>
<221> MISC_FEATURE
<222> (372)..(372)
<223> Wherein amino acid could be Ala
<220>
<221> MISC_FEATURE
<222> (375)..(375)
<223> Wherein amino acid could be Ala
<220>
<221> MISC_FEATURE
<222> (388)..(388)
<223> Wherein amino acid could be Ala
<220>
<221> MISC_FEATURE
<222> (450)..(450)
<223> Wherein amino acid could be Asn
<220>
<221> MISC_FEATURE
<222> (465)..(465)
<223> Wherein amino acid could be Ala
<220>
<221> MISC_FEATURE
<222> (560)..(560)
<223> Wherein amino acid could be Ala
<220>
<221> MISC_FEATURE
<222> (564)..(564)
<223> Wherein amino acid could be Pro
<220>
<221> MISC_FEATURE
<222> (573)..(573)
<223> Wherein amino acid could be Ala
<220>
<221> MISC_FEATURE
<222> (589)..(589)
<223> Wherein amino acid could be Val
<220>
<221> MISC_FEATURE
<222> (592)..(592)
<223> Wherein amino acid could be Gly
<220>
<221> MISC_FEATURE
<222> (594)..(594)
<223> Wherein amino acid could be Leu
<400> 119
Met Gly Ser Ser Leu Asp Asp Glu His Ile Leu Ser Ala Leu Leu Gln
1 5 10 15
Ser Asp Asp Glu Leu Val Gly Glu Asp Ser Asp Ser Glu Val Ser Asp
20 25 30
His Val Ser Glu Asp Asp Val Gln Ser Asp Thr Glu Glu Ala Phe Ile
35 40 45
Asp Glu Val His Glu Val Gln Pro Thr Ser Ser Gly Ser Glu Ile Leu
50 55 60
Asp Glu Gln Asn Val Ile Glu Gln Pro Gly Ser Ser Leu Ala Ser Asn
65 70 75 80
Arg Ile Leu Thr Leu Pro Gln Arg Thr Ile Arg Gly Lys Asn Lys His
85 90 95
Cys Trp Ser Thr Ser Lys Pro Thr Arg Arg Ser Arg Val Ser Ala Leu
100 105 110
Asn Ile Val Arg Ser Gln Arg Gly Pro Thr Arg Met Cys Arg Asn Ile
115 120 125
Tyr Asp Pro Leu Leu Cys Phe Lys Leu Phe Phe Thr Asp Glu Ile Ile
130 135 140
Ser Glu Ile Val Lys Trp Thr Asn Ala Glu Ile Ser Leu Lys Arg Arg
145 150 155 160
Glu Ser Met Thr Ser Ala Thr Phe Arg Asp Thr Asn Glu Asp Glu Ile
165 170 175
Tyr Ala Phe Phe Gly Ile Leu Val Met Thr Ala Val Arg Lys Asp Asn
180 185 190
His Met Ser Thr Asp Asp Leu Phe Asp Arg Ser Leu Ser Met Val Tyr
195 200 205
Val Ser Val Met Ser Arg Asp Arg Phe Asp Phe Leu Ile Arg Cys Leu
210 215 220
Arg Met Asp Asp Lys Ser Ile Arg Pro Thr Leu Arg Glu Asn Asp Val
225 230 235 240
Phe Thr Pro Val Arg Lys Ile Trp Asp Leu Phe Ile His Gln Cys Ile
245 250 255
Gln Asn Tyr Thr Pro Gly Ala His Leu Thr Ile Asp Glu Gln Leu Leu
260 265 270
Gly Phe Arg Gly Arg Cys Pro Phe Arg Val Tyr Ile Pro Asn Lys Pro
275 280 285
Ser Lys Tyr Gly Ile Lys Ile Leu Met Met Cys Asp Ser Gly Thr Lys
290 295 300
Tyr Met Ile Asn Gly Met Pro Tyr Leu Gly Arg Gly Thr Gln Thr Asn
305 310 315 320
Gly Val Pro Leu Gly Glu Tyr Tyr Val Lys Glu Leu Ser Lys Pro Val
325 330 335
His Gly Ser Cys Arg Asn Ile Thr Cys Asp Asn Trp Phe Thr Ser Ile
340 345 350
Pro Leu Ala Lys Asn Leu Leu Gln Glu Pro Tyr Lys Leu Thr Ile Val
355 360 365
Gly Thr Val Arg Ser Asn Lys Arg Glu Ile Pro Glu Val Leu Lys Asn
370 375 380
Ser Arg Ser Arg Pro Val Gly Thr Ser Met Phe Cys Phe Asp Gly Pro
385 390 395 400
Leu Thr Leu Val Ser Tyr Lys Pro Lys Pro Ala Lys Met Val Tyr Leu
405 410 415
Leu Ser Ser Cys Asp Glu Asp Ala Ser Ile Asn Glu Ser Thr Gly Lys
420 425 430
Pro Gln Met Val Met Tyr Tyr Asn Gln Thr Lys Gly Gly Val Asp Thr
435 440 445
Leu Asp Gln Met Cys Ser Val Met Thr Cys Ser Arg Lys Thr Asn Arg
450 455 460
Trp Pro Met Ala Leu Leu Tyr Gly Met Ile Asn Ile Ala Cys Ile Asn
465 470 475 480
Ser Phe Ile Ile Tyr Ser His Asn Val Ser Ser Lys Gly Glu Lys Val
485 490 495
Gln Ser Arg Lys Lys Phe Met Arg Asn Leu Tyr Met Gly Leu Thr Ser
500 505 510
Ser Phe Met Arg Lys Arg Leu Glu Ala Pro Thr Leu Lys Arg Tyr Leu
515 520 525
Arg Asp Asn Ile Ser Asn Ile Leu Pro Lys Glu Val Pro Gly Thr Ser
530 535 540
Asp Asp Ser Thr Glu Glu Pro Val Met Lys Lys Arg Thr Tyr Cys Thr
545 550 555 560
Tyr Cys Pro Ser Lys Ile Arg Arg Lys Ala Ser Ala Ser Cys Lys Lys
565 570 575
Cys Lys Lys Val Ile Cys Arg Glu His Asn Ile Asp Met Cys Gln Ser
580 585 590
Cys Phe
<210> 120
<211> 594
<212> PRT
<213> Artificial Sequence
<220>
<223> Top1 Modified hyperactive PiggyBac aa sequence
<220>
<221> MISC_FEATURE
<222> (245)..(245)
<223> Wherein amino acid could be Ala
<220>
<221> MISC_FEATURE
<222> (275)..(275)
<223> Wherein amino acid could be Arg, Ala
<220>
<221> MISC_FEATURE
<222> (277)..(277)
<223> Wherein amino acid could be Arg, Ala
<220>
<221> MISC_FEATURE
<222> (325)..(325)
<223> Wherein amino acid could be Ala, Gly
<220>
<221> MISC_FEATURE
<222> (347)..(347)
<223> Wherein amino acid could be Asn, Ala
<220>
<221> MISC_FEATURE
<222> (351)..(351)
<223> Wherein amino acid could be Glu, Pro, Ala
<220>
<221> MISC_FEATURE
<222> (372)..(372)
<223> Wherein amino acid could be Arg
<400> 120
Met Gly Ser Ser Leu Asp Asp Glu His Ile Leu Ser Ala Leu Leu Gln
1 5 10 15
Ser Asp Asp Glu Leu Val Gly Glu Asp Ser Asp Ser Glu Val Ser Asp
20 25 30
His Val Ser Glu Asp Asp Val Gln Ser Asp Thr Glu Glu Ala Phe Ile
35 40 45
Asp Glu Val His Glu Val Gln Pro Thr Ser Ser Gly Ser Glu Ile Leu
50 55 60
Asp Glu Gln Asn Val Ile Glu Gln Pro Gly Ser Ser Leu Ala Ser Asn
65 70 75 80
Arg Ile Leu Thr Leu Pro Gln Arg Thr Ile Arg Gly Lys Asn Lys His
85 90 95
Cys Trp Ser Thr Ser Lys Pro Thr Arg Arg Ser Arg Val Ser Ala Leu
100 105 110
Asn Ile Val Arg Ser Gln Arg Gly Pro Thr Arg Met Cys Arg Asn Ile
115 120 125
Tyr Asp Pro Leu Leu Cys Phe Lys Leu Phe Phe Thr Asp Glu Ile Ile
130 135 140
Ser Glu Ile Val Lys Trp Thr Asn Ala Glu Ile Ser Leu Lys Arg Arg
145 150 155 160
Glu Ser Met Thr Ser Ala Thr Phe Arg Asp Thr Asn Glu Asp Glu Ile
165 170 175
Tyr Ala Phe Phe Gly Ile Leu Val Met Thr Ala Val Arg Lys Asp Asn
180 185 190
His Met Ser Thr Asp Asp Leu Phe Asp Arg Ser Leu Ser Met Val Tyr
195 200 205
Val Ser Val Met Ser Arg Asp Arg Phe Asp Phe Leu Ile Arg Cys Leu
210 215 220
Arg Met Asp Asp Lys Ser Ile Arg Pro Thr Leu Arg Glu Asn Asp Val
225 230 235 240
Phe Thr Pro Val Arg Lys Ile Trp Asp Leu Phe Ile His Gln Cys Ile
245 250 255
Gln Asn Tyr Thr Pro Gly Ala His Leu Thr Ile Asp Glu Gln Leu Leu
260 265 270
Gly Phe Arg Gly Arg Cys Pro Phe Arg Val Tyr Ile Pro Asn Lys Pro
275 280 285
Ser Lys Tyr Gly Ile Lys Ile Leu Met Met Cys Asp Ser Gly Thr Lys
290 295 300
Tyr Met Ile Asn Gly Met Pro Tyr Leu Gly Arg Gly Thr Gln Thr Asn
305 310 315 320
Gly Val Pro Leu Gly Glu Tyr Tyr Val Lys Glu Leu Ser Lys Pro Val
325 330 335
His Gly Ser Cys Arg Asn Ile Thr Cys Asp Asn Trp Phe Thr Ser Ile
340 345 350
Pro Leu Ala Lys Asn Leu Leu Gln Glu Pro Tyr Lys Leu Thr Ile Val
355 360 365
Gly Thr Val Arg Ser Asn Lys Arg Glu Ile Pro Glu Val Leu Lys Asn
370 375 380
Ser Arg Ser Arg Pro Val Gly Thr Ser Met Phe Cys Phe Asp Gly Pro
385 390 395 400
Leu Thr Leu Val Ser Tyr Lys Pro Lys Pro Ala Lys Met Val Tyr Leu
405 410 415
Leu Ser Ser Cys Asp Glu Asp Ala Ser Ile Asn Glu Ser Thr Gly Lys
420 425 430
Pro Gln Met Val Met Tyr Tyr Asn Gln Thr Lys Gly Gly Val Asp Thr
435 440 445
Leu Asp Gln Met Cys Ser Val Met Thr Cys Ser Arg Lys Thr Asn Arg
450 455 460
Trp Pro Met Ala Leu Leu Tyr Gly Met Ile Asn Ile Ala Cys Ile Asn
465 470 475 480
Ser Phe Ile Ile Tyr Ser His Asn Val Ser Ser Lys Gly Glu Lys Val
485 490 495
Gln Ser Arg Lys Lys Phe Met Arg Asn Leu Tyr Met Gly Leu Thr Ser
500 505 510
Ser Phe Met Arg Lys Arg Leu Glu Ala Pro Thr Leu Lys Arg Tyr Leu
515 520 525
Arg Asp Asn Ile Ser Asn Ile Leu Pro Lys Glu Val Pro Gly Thr Ser
530 535 540
Asp Asp Ser Thr Glu Glu Pro Val Met Lys Lys Arg Thr Tyr Cys Thr
545 550 555 560
Tyr Cys Pro Ser Lys Ile Arg Arg Lys Ala Ser Ala Ser Cys Lys Lys
565 570 575
Cys Lys Lys Val Ile Cys Arg Glu His Asn Ile Asp Met Cys Gln Ser
580 585 590
Cys Phe
<210> 121
<211> 594
<212> PRT
<213> Artificial Sequence
<220>
<223> Top1.1 Modified hyperactive PiggyBac aa sequence
<220>
<221> MISC_FEATURE
<222> (450)..(450)
<223> Wherein the amino acid can be Asp, Asn
<220>
<221> MISC_FEATURE
<222> (456)..(456)
<223> Wherein the amino acid can be Trp, Ala
<400> 121
Met Gly Ser Ser Leu Asp Asp Glu His Ile Leu Ser Ala Leu Leu Gln
1 5 10 15
Ser Asp Asp Glu Leu Val Gly Glu Asp Ser Asp Ser Glu Val Ser Asp
20 25 30
His Val Ser Glu Asp Asp Val Gln Ser Asp Thr Glu Glu Ala Phe Ile
35 40 45
Asp Glu Val His Glu Val Gln Pro Thr Ser Ser Gly Ser Glu Ile Leu
50 55 60
Asp Glu Gln Asn Val Ile Glu Gln Pro Gly Ser Ser Leu Ala Ser Asn
65 70 75 80
Arg Ile Leu Thr Leu Pro Gln Arg Thr Ile Arg Gly Lys Asn Lys His
85 90 95
Cys Trp Ser Thr Ser Lys Pro Thr Arg Arg Ser Arg Val Ser Ala Leu
100 105 110
Asn Ile Val Arg Ser Gln Arg Gly Pro Thr Arg Met Cys Arg Asn Ile
115 120 125
Tyr Asp Pro Leu Leu Cys Phe Lys Leu Phe Phe Thr Asp Glu Ile Ile
130 135 140
Ser Glu Ile Val Lys Trp Thr Asn Ala Glu Ile Ser Leu Lys Arg Arg
145 150 155 160
Glu Ser Met Thr Ser Ala Thr Phe Arg Asp Thr Asn Glu Asp Glu Ile
165 170 175
Tyr Ala Phe Phe Gly Ile Leu Val Met Thr Ala Val Arg Lys Asp Asn
180 185 190
His Met Ser Thr Asp Asp Leu Phe Asp Arg Ser Leu Ser Met Val Tyr
195 200 205
Val Ser Val Met Ser Arg Asp Arg Phe Asp Phe Leu Ile Arg Cys Leu
210 215 220
Arg Met Asp Asp Lys Ser Ile Arg Pro Thr Leu Arg Glu Asn Asp Val
225 230 235 240
Phe Thr Pro Val Ala Lys Ile Trp Asp Leu Phe Ile His Gln Cys Ile
245 250 255
Gln Asn Tyr Thr Pro Gly Ala His Leu Thr Ile Asp Glu Gln Leu Leu
260 265 270
Gly Phe Arg Gly Arg Cys Pro Phe Arg Val Tyr Ile Pro Asn Lys Pro
275 280 285
Ser Lys Tyr Gly Ile Lys Ile Leu Met Met Cys Asp Ser Gly Thr Lys
290 295 300
Tyr Met Ile Asn Gly Met Pro Tyr Leu Gly Arg Gly Thr Gln Thr Asn
305 310 315 320
Gly Val Pro Leu Ala Glu Tyr Tyr Val Lys Glu Leu Ser Lys Pro Val
325 330 335
His Gly Ser Cys Arg Asn Ile Thr Cys Asp Asn Trp Phe Thr Glu Ile
340 345 350
Pro Leu Ala Lys Asn Leu Leu Gln Glu Pro Tyr Lys Leu Thr Ile Val
355 360 365
Gly Thr Val Arg Ser Asn Ala Arg Glu Ile Pro Glu Val Leu Lys Asn
370 375 380
Ser Arg Ser Arg Pro Val Gly Thr Ser Met Phe Cys Phe Asp Gly Pro
385 390 395 400
Leu Thr Leu Val Ser Tyr Lys Pro Lys Pro Ala Lys Met Val Tyr Leu
405 410 415
Leu Ser Ser Cys Asp Glu Asp Ala Ser Ile Asn Glu Ser Thr Gly Lys
420 425 430
Pro Gln Met Val Met Tyr Tyr Asn Gln Thr Lys Gly Gly Val Asp Thr
435 440 445
Leu Asp Gln Met Cys Ser Val Met Thr Cys Ser Arg Lys Thr Asn Arg
450 455 460
Trp Pro Met Ala Leu Leu Tyr Gly Met Ile Asn Ile Ala Cys Ile Asn
465 470 475 480
Ser Phe Ile Ile Tyr Ser His Asn Val Ser Ser Lys Gly Glu Lys Val
485 490 495
Gln Ser Arg Lys Lys Phe Met Arg Asn Leu Tyr Met Gly Leu Thr Ser
500 505 510
Ser Phe Met Arg Lys Arg Leu Glu Ala Pro Thr Leu Lys Arg Tyr Leu
515 520 525
Arg Asp Asn Ile Ser Asn Ile Leu Pro Lys Glu Val Pro Gly Thr Ser
530 535 540
Asp Asp Ser Thr Glu Glu Pro Val Met Lys Lys Arg Thr Tyr Cys Thr
545 550 555 560
Tyr Cys Pro Pro Lys Ile Arg Arg Lys Ala Ser Ala Ser Cys Lys Lys
565 570 575
Cys Lys Lys Val Ile Cys Arg Glu His Asn Ile Asp Met Cys Gln Gly
580 585 590
Cys Leu
<210> 122
<211> 594
<212> PRT
<213> Artificial Sequence
<220>
<223> Top1.2 Modified hyperactive PiggyBac aa sequence
<220>
<221> MISC_FEATURE
<222> (450)..(450)
<223> Wherein the amino acid can be Asp, Asn
<220>
<221> MISC_FEATURE
<222> (456)..(456)
<223> Wherein the amino acid can be Trp, Ala
<400> 122
Met Gly Ser Ser Leu Asp Asp Glu His Ile Leu Ser Ala Leu Leu Gln
1 5 10 15
Ser Asp Asp Glu Leu Val Gly Glu Asp Ser Asp Ser Glu Val Ser Asp
20 25 30
His Val Ser Glu Asp Asp Val Gln Ser Asp Thr Glu Glu Ala Phe Ile
35 40 45
Asp Glu Val His Glu Val Gln Pro Thr Ser Ser Gly Ser Glu Ile Leu
50 55 60
Asp Glu Gln Asn Val Ile Glu Gln Pro Gly Ser Ser Leu Ala Ser Asn
65 70 75 80
Arg Ile Leu Thr Leu Pro Gln Arg Thr Ile Arg Gly Lys Asn Lys His
85 90 95
Cys Trp Ser Thr Ser Lys Pro Thr Arg Arg Ser Arg Val Ser Ala Leu
100 105 110
Asn Ile Val Arg Ser Gln Arg Gly Pro Thr Arg Met Cys Arg Asn Ile
115 120 125
Tyr Asp Pro Leu Leu Cys Phe Lys Leu Phe Phe Thr Asp Glu Ile Ile
130 135 140
Ser Glu Ile Val Lys Trp Thr Asn Ala Glu Ile Ser Leu Lys Arg Arg
145 150 155 160
Glu Ser Met Thr Ser Ala Thr Phe Arg Asp Thr Asn Glu Asp Glu Ile
165 170 175
Tyr Ala Phe Phe Gly Ile Leu Val Met Thr Ala Val Arg Lys Asp Asn
180 185 190
His Met Ser Thr Asp Asp Leu Phe Asp Arg Ser Leu Ser Met Val Tyr
195 200 205
Val Ser Val Met Ser Arg Asp Arg Phe Asp Phe Leu Ile Arg Cys Leu
210 215 220
Arg Met Asp Asp Lys Ser Ile Arg Pro Thr Leu Arg Glu Asn Asp Val
225 230 235 240
Phe Thr Pro Val Ala Lys Ile Trp Asp Leu Phe Ile His Gln Cys Ile
245 250 255
Gln Asn Tyr Thr Pro Gly Ala His Leu Thr Ile Asp Glu Gln Leu Leu
260 265 270
Gly Phe Ala Gly Ala Cys Pro Phe Arg Val Tyr Ile Pro Asn Lys Pro
275 280 285
Ser Lys Tyr Gly Ile Lys Ile Leu Met Met Cys Asp Ser Gly Thr Lys
290 295 300
Tyr Met Ile Asn Gly Met Pro Tyr Leu Gly Arg Gly Thr Gln Thr Asn
305 310 315 320
Gly Val Pro Leu Gly Glu Tyr Tyr Val Lys Glu Leu Ser Lys Pro Val
325 330 335
His Gly Ser Cys Arg Asn Ile Thr Cys Asp Ala Trp Phe Thr Pro Ile
340 345 350
Pro Leu Ala Lys Asn Leu Leu Gln Glu Pro Tyr Lys Leu Thr Ile Val
355 360 365
Gly Thr Val Arg Ser Asn Ala Arg Glu Ile Pro Glu Val Leu Lys Asn
370 375 380
Ser Arg Ser Arg Pro Val Gly Thr Ser Met Phe Cys Phe Asp Gly Pro
385 390 395 400
Leu Thr Leu Val Ser Tyr Lys Pro Lys Pro Ala Lys Met Val Tyr Leu
405 410 415
Leu Ser Ser Cys Asp Glu Asp Ala Ser Ile Asn Glu Ser Thr Gly Lys
420 425 430
Pro Gln Met Val Met Tyr Tyr Asn Gln Thr Lys Gly Gly Val Asp Thr
435 440 445
Leu Asp Gln Met Cys Ser Val Met Thr Cys Ser Arg Lys Thr Asn Arg
450 455 460
Trp Pro Met Ala Leu Leu Tyr Gly Met Ile Asn Ile Ala Cys Ile Asn
465 470 475 480
Ser Phe Ile Ile Tyr Ser His Asn Val Ser Ser Lys Gly Glu Lys Val
485 490 495
Gln Ser Arg Lys Lys Phe Met Arg Asn Leu Tyr Met Gly Leu Thr Ser
500 505 510
Ser Phe Met Arg Lys Arg Leu Glu Ala Pro Thr Leu Lys Arg Tyr Leu
515 520 525
Arg Asp Asn Ile Ser Asn Ile Leu Pro Lys Glu Val Pro Gly Thr Ser
530 535 540
Asp Asp Ser Thr Glu Glu Pro Val Met Lys Lys Arg Thr Tyr Cys Ala
545 550 555 560
Tyr Cys Pro Ser Lys Ile Arg Arg Lys Ala Ser Ala Ser Cys Lys Lys
565 570 575
Cys Lys Lys Val Ile Cys Arg Glu His Asn Ile Asp Met Cys Gln Ser
580 585 590
Cys Phe
<210> 123
<211> 594
<212> PRT
<213> Artificial Sequence
<220>
<223> Top1.3 Modified hyperactive PiggyBac aa sequence
<220>
<221> MISC_FEATURE
<222> (450)..(450)
<223> Wherein the amino acid can be Asp, Asn
<220>
<221> MISC_FEATURE
<222> (456)..(456)
<223> Wherein the amino acid can be Trp, Ala
<400> 123
Met Gly Ser Ser Leu Asp Asp Glu His Ile Leu Ser Ala Leu Leu Gln
1 5 10 15
Ser Asp Asp Glu Leu Val Gly Glu Asp Ser Asp Ser Glu Val Ser Asp
20 25 30
His Val Ser Glu Asp Asp Val Gln Ser Asp Thr Glu Glu Ala Phe Ile
35 40 45
Asp Glu Val His Glu Val Gln Pro Thr Ser Ser Gly Ser Glu Ile Leu
50 55 60
Asp Glu Gln Asn Val Ile Glu Gln Pro Gly Ser Ser Leu Ala Ser Asn
65 70 75 80
Arg Ile Leu Thr Leu Pro Gln Arg Thr Ile Arg Gly Lys Asn Lys His
85 90 95
Cys Trp Ser Thr Ser Lys Pro Thr Arg Arg Ser Arg Val Ser Ala Leu
100 105 110
Asn Ile Val Arg Ser Gln Arg Gly Pro Thr Arg Met Cys Arg Asn Ile
115 120 125
Tyr Asp Pro Leu Leu Cys Phe Lys Leu Phe Phe Thr Asp Glu Ile Ile
130 135 140
Ser Glu Ile Val Lys Trp Thr Asn Ala Glu Ile Ser Leu Lys Arg Arg
145 150 155 160
Glu Ser Met Thr Ser Ala Thr Phe Arg Asp Thr Asn Glu Asp Glu Ile
165 170 175
Tyr Ala Phe Phe Gly Ile Leu Val Met Thr Ala Val Arg Lys Asp Asn
180 185 190
His Met Ser Thr Asp Asp Leu Phe Asp Arg Ser Leu Ser Met Val Tyr
195 200 205
Val Ser Val Met Ser Arg Asp Arg Phe Asp Phe Leu Ile Arg Cys Leu
210 215 220
Arg Met Asp Asp Lys Ser Ile Arg Pro Thr Leu Arg Glu Asn Asp Val
225 230 235 240
Phe Thr Pro Val Ala Lys Ile Trp Asp Leu Phe Ile His Gln Cys Ile
245 250 255
Gln Asn Tyr Thr Pro Gly Ala His Leu Thr Ile Asp Glu Gln Leu Leu
260 265 270
Gly Phe Arg Gly Arg Cys Pro Phe Arg Val Tyr Ile Pro Asn Lys Pro
275 280 285
Ser Lys Tyr Gly Ile Lys Ile Leu Met Met Cys Asp Ser Gly Thr Lys
290 295 300
Tyr Met Ile Asn Gly Met Pro Tyr Leu Gly Arg Gly Thr Gln Thr Asn
305 310 315 320
Gly Val Pro Leu Ala Glu Tyr Tyr Val Lys Glu Leu Ser Lys Pro Val
325 330 335
His Gly Ser Cys Arg Asn Ile Thr Cys Asp Ala Trp Phe Thr Ala Ile
340 345 350
Pro Leu Ala Lys Asn Leu Leu Gln Glu Pro Tyr Lys Leu Thr Ile Val
355 360 365
Gly Thr Val Arg Ser Asn Ala Arg Glu Ile Pro Glu Val Leu Lys Asn
370 375 380
Ser Arg Ser Arg Pro Val Gly Thr Ser Met Phe Cys Phe Asp Gly Pro
385 390 395 400
Leu Thr Leu Val Ser Tyr Lys Pro Lys Pro Ala Lys Met Val Tyr Leu
405 410 415
Leu Ser Ser Cys Asp Glu Asp Ala Ser Ile Asn Glu Ser Thr Gly Lys
420 425 430
Pro Gln Met Val Met Tyr Tyr Asn Gln Thr Lys Gly Gly Val Asp Thr
435 440 445
Leu Asp Gln Met Cys Ser Val Met Thr Cys Ser Arg Lys Thr Asn Arg
450 455 460
Trp Pro Met Ala Leu Leu Tyr Gly Met Ile Asn Ile Ala Cys Ile Asn
465 470 475 480
Ser Phe Ile Ile Tyr Ser His Asn Val Ser Ser Lys Gly Glu Lys Val
485 490 495
Gln Ser Arg Lys Lys Phe Met Arg Asn Leu Tyr Met Gly Leu Thr Ser
500 505 510
Ser Phe Met Arg Lys Arg Leu Glu Ala Pro Thr Leu Lys Arg Tyr Leu
515 520 525
Arg Asp Asn Ile Ser Asn Ile Leu Pro Lys Glu Val Pro Gly Thr Ser
530 535 540
Asp Asp Ser Thr Glu Glu Pro Val Met Lys Lys Arg Thr Tyr Cys Ala
545 550 555 560
Tyr Cys Pro Ser Lys Ile Arg Arg Lys Ala Ser Ala Ala Cys Lys Lys
565 570 575
Cys Lys Lys Val Ile Cys Arg Glu His Asn Ile Asp Met Cys Gln Ser
580 585 590
Cys Phe
<210> 124
<211> 594
<212> PRT
<213> Artificial Sequence
<220>
<223> Regular modified 1 hyperactive PiggyBac aa sequence
<220>
<221> MISC_FEATURE
<222> (450)..(450)
<223> Wherein the amino acid can be Asp, Asn
<220>
<221> MISC_FEATURE
<222> (456)..(456)
<223> Wherein the amino acid can be Trp, Ala
<400> 124
Met Gly Ser Ser Leu Asp Asp Glu His Ile Leu Ser Ala Leu Leu Gln
1 5 10 15
Ser Asp Asp Glu Leu Val Gly Glu Asp Ser Asp Ser Glu Val Ser Asp
20 25 30
His Val Ser Glu Asp Asp Val Gln Ser Asp Thr Glu Glu Ala Phe Ile
35 40 45
Asp Glu Val His Glu Val Gln Pro Thr Ser Ser Gly Ser Glu Ile Leu
50 55 60
Asp Glu Gln Asn Val Ile Glu Gln Pro Gly Ser Ser Leu Ala Ser Asn
65 70 75 80
Arg Ile Leu Thr Leu Pro Gln Arg Thr Ile Arg Gly Lys Asn Lys His
85 90 95
Cys Trp Ser Thr Ser Lys Pro Thr Arg Arg Ser Arg Val Ser Ala Leu
100 105 110
Asn Ile Val Arg Ser Gln Arg Gly Pro Thr Arg Met Cys Arg Asn Ile
115 120 125
Tyr Asp Pro Leu Leu Cys Phe Lys Leu Phe Phe Thr Asp Glu Ile Ile
130 135 140
Ser Glu Ile Val Lys Trp Thr Asn Ala Glu Ile Ser Leu Lys Arg Arg
145 150 155 160
Glu Ser Met Thr Ser Ala Thr Phe Arg Asp Thr Asn Glu Asp Glu Ile
165 170 175
Tyr Ala Phe Phe Gly Ile Leu Val Met Thr Ala Val Arg Lys Asp Asn
180 185 190
His Met Ser Thr Asp Asp Leu Phe Asp Arg Ser Leu Ser Met Val Tyr
195 200 205
Val Ser Val Met Ser Arg Asp Arg Phe Asp Phe Leu Ile Arg Cys Leu
210 215 220
Arg Met Asp Asp Lys Ser Ile Arg Pro Thr Leu Arg Glu Asn Asp Val
225 230 235 240
Phe Thr Pro Val Arg Lys Ile Trp Asp Leu Phe Ile His Gln Cys Ile
245 250 255
Gln Asn Tyr Thr Pro Gly Ala His Leu Thr Ile Asp Glu Gln Leu Leu
260 265 270
Gly Phe Arg Gly Arg Cys Pro Phe Arg Val Tyr Ile Pro Asn Lys Pro
275 280 285
Ser Lys Tyr Gly Ile Lys Ile Leu Met Met Cys Asp Ser Gly Thr Lys
290 295 300
Tyr Met Ile Asn Gly Met Pro Tyr Leu Gly Arg Gly Thr Gln Thr Asn
305 310 315 320
Gly Val Pro Leu Gly Glu Tyr Tyr Val Lys Glu Leu Ser Lys Pro Val
325 330 335
His Gly Ser Cys Arg Asn Ile Thr Cys Asp Ala Trp Phe Thr Ser Ile
340 345 350
Pro Leu Ala Lys Asn Leu Leu Gln Glu Pro Tyr Lys Leu Thr Ile Val
355 360 365
Gly Thr Val Ala Ser Asn Lys Arg Glu Ile Pro Glu Val Leu Lys Asn
370 375 380
Ser Arg Ser Arg Pro Val Gly Thr Ser Met Phe Cys Phe Asp Gly Pro
385 390 395 400
Leu Thr Leu Val Ser Tyr Lys Pro Lys Pro Ala Lys Met Val Tyr Leu
405 410 415
Leu Ser Ser Cys Asp Glu Asp Ala Ser Ile Asn Glu Ser Thr Gly Lys
420 425 430
Pro Gln Met Val Met Tyr Tyr Asn Gln Thr Lys Gly Gly Val Asp Thr
435 440 445
Leu Asp Gln Met Cys Ser Val Met Thr Cys Ser Arg Lys Thr Asn Arg
450 455 460
Trp Pro Met Ala Leu Leu Tyr Gly Met Ile Asn Ile Ala Cys Ile Asn
465 470 475 480
Ser Phe Ile Ile Tyr Ser His Asn Val Ser Ser Lys Gly Glu Lys Val
485 490 495
Gln Ser Arg Lys Lys Phe Met Arg Asn Leu Tyr Met Gly Leu Thr Ser
500 505 510
Ser Phe Met Arg Lys Arg Leu Glu Ala Pro Thr Leu Lys Arg Tyr Leu
515 520 525
Arg Asp Asn Ile Ser Asn Ile Leu Pro Lys Glu Val Pro Gly Thr Ser
530 535 540
Asp Asp Ser Thr Glu Glu Pro Val Met Lys Lys Arg Thr Tyr Cys Thr
545 550 555 560
Tyr Cys Pro Ser Lys Ile Arg Arg Lys Ala Ser Ala Ser Cys Lys Lys
565 570 575
Cys Lys Lys Val Ile Cys Arg Glu His Asn Ile Asp Met Cys Gln Ser
580 585 590
Cys Phe
<210> 125
<211> 594
<212> PRT
<213> Artificial Sequence
<220>
<223> Regular modified 2 hyperactive PiggyBac aa sequence
<220>
<221> MISC_FEATURE
<222> (450)..(450)
<223> Wherein the amino acid can be Asp, Asn
<220>
<221> MISC_FEATURE
<222> (456)..(456)
<223> Wherein the amino acid can be Trp, Ala
<400> 125
Met Gly Ser Ser Leu Asp Asp Glu His Ile Leu Ser Ala Leu Leu Gln
1 5 10 15
Ser Asp Asp Glu Leu Val Gly Glu Asp Ser Asp Ser Glu Val Ser Asp
20 25 30
His Val Ser Glu Asp Asp Val Gln Ser Asp Thr Glu Glu Ala Phe Ile
35 40 45
Asp Glu Val His Glu Val Gln Pro Thr Ser Ser Gly Ser Glu Ile Leu
50 55 60
Asp Glu Gln Asn Val Ile Glu Gln Pro Gly Ser Ser Leu Ala Ser Asn
65 70 75 80
Arg Ile Leu Thr Leu Pro Gln Arg Thr Ile Arg Gly Lys Asn Lys His
85 90 95
Cys Trp Ser Thr Ser Lys Pro Thr Arg Arg Ser Arg Val Ser Ala Leu
100 105 110
Asn Ile Val Arg Ser Gln Arg Gly Pro Thr Arg Met Cys Arg Asn Ile
115 120 125
Tyr Asp Pro Leu Leu Cys Phe Lys Leu Phe Phe Thr Asp Glu Ile Ile
130 135 140
Ser Glu Ile Val Lys Trp Thr Asn Ala Glu Ile Ser Leu Lys Arg Arg
145 150 155 160
Glu Ser Met Thr Ser Ala Thr Phe Arg Asp Thr Asn Glu Asp Glu Ile
165 170 175
Tyr Ala Phe Phe Gly Ile Leu Val Met Thr Ala Val Arg Lys Asp Asn
180 185 190
His Met Ser Thr Asp Asp Leu Phe Asp Arg Ser Leu Ser Met Val Tyr
195 200 205
Val Ser Val Met Ser Arg Asp Arg Phe Asp Phe Leu Ile Arg Cys Leu
210 215 220
Arg Met Asp Asp Lys Ser Ile Arg Pro Thr Leu Arg Glu Asn Asp Val
225 230 235 240
Phe Thr Pro Val Arg Lys Ile Trp Asp Leu Phe Ile His Gln Cys Ile
245 250 255
Gln Asn Tyr Thr Pro Gly Ala His Leu Thr Ile Asp Glu Gln Leu Leu
260 265 270
Gly Phe Arg Gly Arg Cys Pro Phe Arg Val Tyr Ile Pro Asn Lys Pro
275 280 285
Ser Lys Tyr Gly Ile Lys Ile Leu Met Met Cys Asp Ser Gly Thr Lys
290 295 300
Tyr Met Ile Asn Gly Met Pro Tyr Leu Gly Arg Gly Thr Gln Thr Asn
305 310 315 320
Gly Val Pro Leu Gly Glu Tyr Tyr Val Lys Glu Leu Ser Lys Pro Val
325 330 335
His Gly Ser Cys Arg Asn Ile Thr Cys Asp Asn Trp Phe Thr Ser Ile
340 345 350
Pro Leu Ala Lys Asn Leu Leu Gln Glu Pro Tyr Lys Leu Thr Ile Val
355 360 365
Gly Thr Val Arg Ser Asn Lys Arg Glu Ile Pro Glu Val Leu Lys Asn
370 375 380
Ser Arg Ser Arg Pro Val Gly Thr Ser Met Phe Cys Phe Asp Gly Pro
385 390 395 400
Leu Thr Leu Val Ser Tyr Lys Pro Lys Pro Ala Lys Met Val Tyr Leu
405 410 415
Leu Ser Ser Cys Asp Glu Asp Ala Ser Ile Asn Glu Ser Thr Gly Lys
420 425 430
Pro Gln Met Val Met Tyr Tyr Asn Gln Thr Lys Gly Gly Val Asp Thr
435 440 445
Leu Asp Gln Met Cys Ser Val Met Thr Cys Ser Arg Lys Thr Asn Arg
450 455 460
Trp Pro Met Ala Leu Leu Tyr Gly Met Ile Asn Ile Ala Cys Ile Asn
465 470 475 480
Ser Phe Ile Ile Tyr Ser His Asn Val Ser Ser Lys Gly Glu Lys Val
485 490 495
Gln Ser Arg Lys Lys Phe Met Arg Asn Leu Tyr Met Gly Leu Thr Ser
500 505 510
Ser Phe Met Arg Lys Arg Leu Glu Ala Pro Thr Leu Lys Arg Tyr Leu
515 520 525
Arg Asp Asn Ile Ser Asn Ile Leu Pro Lys Glu Val Pro Gly Thr Ser
530 535 540
Asp Asp Ser Thr Glu Glu Pro Val Met Lys Lys Arg Thr Tyr Cys Thr
545 550 555 560
Tyr Cys Pro Ser Lys Ile Arg Arg Lys Ala Ser Ala Ser Cys Lys Lys
565 570 575
Cys Lys Lys Val Ile Cys Arg Glu His Asn Ile Asp Met Cys Gln Gly
580 585 590
Cys Phe
<210> 126
<211> 594
<212> PRT
<213> Artificial Sequence
<220>
<223> Regular modified 3 hyperactive PiggyBac aa sequence
<220>
<221> MISC_FEATURE
<222> (450)..(450)
<223> Wherein the amino acid can be Asp, Asn
<220>
<221> MISC_FEATURE
<222> (456)..(456)
<223> Wherein the amino acid can be Trp, Ala
<400> 126
Met Gly Ser Ser Leu Asp Asp Glu His Ile Leu Ser Ala Leu Leu Gln
1 5 10 15
Ser Asp Asp Glu Leu Val Gly Glu Asp Ser Asp Ser Glu Val Ser Asp
20 25 30
His Val Ser Glu Asp Asp Val Gln Ser Asp Thr Glu Glu Ala Phe Ile
35 40 45
Asp Glu Val His Glu Val Gln Pro Thr Ser Ser Gly Ser Glu Ile Leu
50 55 60
Asp Glu Gln Asn Val Ile Glu Gln Pro Gly Ser Ser Leu Ala Ser Asn
65 70 75 80
Arg Ile Leu Thr Leu Pro Gln Arg Thr Ile Arg Gly Lys Asn Lys His
85 90 95
Cys Trp Ser Thr Ser Lys Pro Thr Arg Arg Ser Arg Val Ser Ala Leu
100 105 110
Asn Ile Val Arg Ser Gln Arg Gly Pro Thr Arg Met Cys Arg Asn Ile
115 120 125
Tyr Asp Pro Leu Leu Cys Phe Lys Leu Phe Phe Thr Asp Glu Ile Ile
130 135 140
Ser Glu Ile Val Lys Trp Thr Asn Ala Glu Ile Ser Leu Lys Arg Arg
145 150 155 160
Glu Ser Met Thr Ser Ala Thr Phe Arg Asp Thr Asn Glu Asp Glu Ile
165 170 175
Tyr Ala Phe Phe Gly Ile Leu Val Met Thr Ala Val Arg Lys Asp Asn
180 185 190
His Met Ser Thr Asp Asp Leu Phe Asp Arg Ser Leu Ser Met Val Tyr
195 200 205
Val Ser Val Met Ser Arg Asp Arg Phe Asp Phe Leu Ile Arg Cys Leu
210 215 220
Arg Met Asp Asp Lys Ser Ile Arg Pro Thr Leu Arg Glu Asn Asp Val
225 230 235 240
Phe Thr Pro Val Arg Lys Ile Trp Asp Leu Phe Ile His Gln Cys Ile
245 250 255
Gln Asn Tyr Thr Pro Gly Ala His Leu Thr Ile Asp Glu Gln Leu Leu
260 265 270
Gly Phe Arg Gly Arg Cys Pro Phe Arg Val Tyr Ile Pro Asn Lys Pro
275 280 285
Ser Lys Tyr Gly Ile Lys Ile Leu Met Met Cys Asp Ser Gly Thr Lys
290 295 300
Tyr Met Ile Asn Gly Met Pro Tyr Leu Gly Arg Gly Thr Gln Thr Asn
305 310 315 320
Gly Val Pro Leu Gly Glu Tyr Tyr Val Lys Glu Leu Ser Lys Pro Val
325 330 335
His Gly Ser Cys Arg Asn Ile Thr Cys Asp Asn Trp Phe Thr Ser Ile
340 345 350
Pro Leu Ala Lys Asn Leu Leu Gln Glu Pro Tyr Lys Leu Thr Ile Val
355 360 365
Gly Thr Val Arg Ser Asn Lys Arg Glu Ile Pro Glu Val Leu Lys Asn
370 375 380
Ser Arg Ser Arg Pro Val Gly Thr Ser Met Phe Cys Phe Asp Gly Pro
385 390 395 400
Leu Thr Leu Val Ser Tyr Lys Pro Lys Pro Ala Lys Met Val Tyr Leu
405 410 415
Leu Ser Ser Cys Asp Glu Asp Ala Ser Ile Asn Glu Ser Thr Gly Lys
420 425 430
Pro Gln Met Val Met Tyr Tyr Asn Gln Thr Lys Gly Gly Val Asp Thr
435 440 445
Leu Asp Gln Met Cys Ser Val Met Thr Cys Ser Arg Lys Thr Asn Arg
450 455 460
Trp Pro Met Ala Leu Leu Tyr Gly Met Ile Asn Ile Ala Cys Ile Asn
465 470 475 480
Ser Phe Ile Ile Tyr Ser His Asn Val Ser Ser Lys Gly Glu Lys Val
485 490 495
Gln Ser Arg Lys Lys Phe Met Arg Asn Leu Tyr Met Gly Leu Thr Ser
500 505 510
Ser Phe Met Arg Lys Arg Leu Glu Ala Pro Thr Leu Lys Arg Tyr Leu
515 520 525
Arg Asp Asn Ile Ser Asn Ile Leu Pro Lys Glu Val Pro Gly Thr Ser
530 535 540
Asp Asp Ser Thr Glu Glu Pro Val Met Lys Lys Arg Thr Tyr Cys Thr
545 550 555 560
Tyr Cys Pro Ser Lys Ile Arg Arg Lys Ala Ser Ala Ser Cys Lys Lys
565 570 575
Cys Lys Lys Val Ile Cys Arg Glu His Asn Ile Asp Met Cys Gln Ser
580 585 590
Cys Phe
<210> 127
<211> 594
<212> PRT
<213> Artificial Sequence
<220>
<223> Regular modified 4 hyperactive PiggyBac aa sequence
<220>
<221> MISC_FEATURE
<222> (450)..(450)
<223> Wherein the amino acid can be Asp, Asn
<220>
<221> MISC_FEATURE
<222> (456)..(456)
<223> Wherein the amino acid can be Trp, Ala
<400> 127
Met Gly Ser Ser Leu Asp Asp Glu His Ile Leu Ser Ala Leu Leu Gln
1 5 10 15
Ser Asp Asp Glu Leu Val Gly Glu Asp Ser Asp Ser Glu Val Ser Asp
20 25 30
His Val Ser Glu Asp Asp Val Gln Ser Asp Thr Glu Glu Ala Phe Ile
35 40 45
Asp Glu Val His Glu Val Gln Pro Thr Ser Ser Gly Ser Glu Ile Leu
50 55 60
Asp Glu Gln Asn Val Ile Glu Gln Pro Gly Ser Ser Leu Ala Ser Asn
65 70 75 80
Arg Ile Leu Thr Leu Pro Gln Arg Thr Ile Arg Gly Lys Asn Lys His
85 90 95
Cys Trp Ser Thr Ser Lys Pro Thr Arg Arg Ser Arg Val Ser Ala Leu
100 105 110
Asn Ile Val Arg Ser Gln Arg Gly Pro Thr Arg Met Cys Arg Asn Ile
115 120 125
Tyr Asp Pro Leu Leu Cys Phe Lys Leu Phe Phe Thr Asp Glu Ile Ile
130 135 140
Ser Glu Ile Val Lys Trp Thr Asn Ala Glu Ile Ser Leu Lys Arg Arg
145 150 155 160
Glu Ser Met Thr Ser Ala Thr Phe Arg Asp Thr Asn Glu Asp Glu Ile
165 170 175
Tyr Ala Phe Phe Gly Ile Leu Val Met Thr Ala Val Arg Lys Asp Asn
180 185 190
His Met Ser Thr Asp Asp Leu Phe Asp Arg Ser Leu Ser Met Val Tyr
195 200 205
Val Ser Val Met Ser Arg Asp Arg Phe Asp Phe Leu Ile Arg Cys Leu
210 215 220
Arg Met Asp Asp Lys Ser Ile Arg Pro Thr Leu Arg Glu Asn Asp Val
225 230 235 240
Phe Thr Pro Val Ala Lys Ile Trp Asp Leu Phe Ile His Gln Cys Ile
245 250 255
Gln Asn Tyr Thr Pro Gly Ala His Leu Thr Ile Asp Glu Gln Leu Leu
260 265 270
Gly Phe Ala Gly Ala Cys Pro Phe Arg Val Tyr Ile Pro Asn Lys Pro
275 280 285
Ser Lys Tyr Gly Ile Lys Ile Leu Met Met Cys Asp Ser Gly Thr Lys
290 295 300
Tyr Met Ile Asn Gly Met Pro Tyr Leu Gly Arg Gly Thr Gln Thr Asn
305 310 315 320
Gly Val Pro Leu Gly Glu Tyr Tyr Val Lys Glu Leu Ser Lys Pro Val
325 330 335
His Gly Ser Cys Arg Asn Ile Thr Cys Asp Asn Trp Phe Thr Ser Ile
340 345 350
Pro Leu Ala Lys Asn Leu Leu Gln Glu Pro Tyr Lys Leu Thr Ile Val
355 360 365
Gly Thr Val Ala Ser Asn Ala Arg Glu Ile Pro Glu Val Leu Lys Asn
370 375 380
Ser Arg Ser Arg Pro Val Gly Thr Ser Met Phe Cys Phe Asp Gly Pro
385 390 395 400
Leu Thr Leu Val Ser Tyr Lys Pro Lys Pro Ala Lys Met Val Tyr Leu
405 410 415
Leu Ser Ser Cys Asp Glu Asp Ala Ser Ile Asn Glu Ser Thr Gly Lys
420 425 430
Pro Gln Met Val Met Tyr Tyr Asn Gln Thr Lys Gly Gly Val Asp Thr
435 440 445
Leu Asp Gln Met Cys Ser Val Met Thr Cys Ser Arg Lys Thr Asn Arg
450 455 460
Trp Pro Met Ala Leu Leu Tyr Gly Met Ile Asn Ile Ala Cys Ile Asn
465 470 475 480
Ser Phe Ile Ile Tyr Ser His Asn Val Ser Ser Lys Gly Glu Lys Val
485 490 495
Gln Ser Arg Lys Lys Phe Met Arg Asn Leu Tyr Met Gly Leu Thr Ser
500 505 510
Ser Phe Met Arg Lys Arg Leu Glu Ala Pro Thr Leu Lys Arg Tyr Leu
515 520 525
Arg Asp Asn Ile Ser Asn Ile Leu Pro Lys Glu Val Pro Gly Thr Ser
530 535 540
Asp Asp Ser Thr Glu Glu Pro Val Met Lys Lys Arg Thr Tyr Cys Thr
545 550 555 560
Tyr Cys Pro Ser Lys Ile Arg Arg Lys Ala Ser Ala Ser Cys Lys Lys
565 570 575
Cys Lys Lys Val Ile Cys Arg Glu His Asn Ile Asp Met Cys Gln Ser
580 585 590
Cys Leu
<210> 128
<211> 594
<212> PRT
<213> Artificial Sequence
<220>
<223> Regular modified 5 hyperactive PiggyBac aa sequence
<220>
<221> MISC_FEATURE
<222> (450)..(450)
<223> Wherein the amino acid can be Asp, Asn
<220>
<221> MISC_FEATURE
<222> (456)..(456)
<223> Wherein the amino acid can be Trp, Ala
<400> 128
Met Gly Ser Ser Leu Asp Asp Glu His Ile Leu Ser Ala Leu Leu Gln
1 5 10 15
Ser Asp Asp Glu Leu Val Gly Glu Asp Ser Asp Ser Glu Val Ser Asp
20 25 30
His Val Ser Glu Asp Asp Val Gln Ser Asp Thr Glu Glu Ala Phe Ile
35 40 45
Asp Glu Val His Glu Val Gln Pro Thr Ser Ser Gly Ser Glu Ile Leu
50 55 60
Asp Glu Gln Asn Val Ile Glu Gln Pro Gly Ser Ser Leu Ala Ser Asn
65 70 75 80
Arg Ile Leu Thr Leu Pro Gln Arg Thr Ile Arg Gly Lys Asn Lys His
85 90 95
Cys Trp Ser Thr Ser Lys Pro Thr Arg Arg Ser Arg Val Ser Ala Leu
100 105 110
Asn Ile Val Arg Ser Gln Arg Gly Pro Thr Arg Met Cys Arg Asn Ile
115 120 125
Tyr Asp Pro Leu Leu Cys Phe Lys Leu Phe Phe Thr Asp Glu Ile Ile
130 135 140
Ser Glu Ile Val Lys Trp Thr Asn Ala Glu Ile Ser Leu Lys Arg Arg
145 150 155 160
Glu Ser Met Thr Ser Ala Thr Phe Arg Asp Thr Asn Glu Asp Glu Ile
165 170 175
Tyr Ala Phe Phe Gly Ile Leu Val Met Thr Ala Val Arg Lys Asp Asn
180 185 190
His Met Ser Thr Asp Asp Leu Phe Asp Arg Ser Leu Ser Met Val Tyr
195 200 205
Val Ser Val Met Ser Arg Asp Arg Phe Asp Phe Leu Ile Arg Cys Leu
210 215 220
Arg Met Asp Asp Lys Ser Ile Arg Pro Thr Leu Arg Glu Asn Asp Val
225 230 235 240
Phe Thr Pro Val Arg Lys Ile Trp Asp Leu Phe Ile His Gln Cys Ile
245 250 255
Gln Asn Tyr Thr Pro Gly Ala His Leu Thr Ile Asp Glu Gln Leu Leu
260 265 270
Gly Phe Ala Gly Arg Cys Pro Phe Arg Val Tyr Ile Pro Asn Lys Pro
275 280 285
Ser Lys Tyr Gly Ile Lys Ile Leu Met Met Cys Asp Ser Gly Thr Lys
290 295 300
Tyr Met Ile Asn Gly Met Pro Tyr Leu Gly Arg Gly Thr Gln Thr Asn
305 310 315 320
Gly Val Pro Leu Ala Glu Tyr Tyr Val Lys Glu Leu Ser Lys Pro Val
325 330 335
His Gly Ser Cys Arg Asn Ile Thr Cys Asp Ser Trp Phe Thr Ala Ile
340 345 350
Pro Leu Ala Lys Asn Leu Leu Gln Glu Pro Tyr Lys Leu Thr Ile Val
355 360 365
Gly Thr Val Ala Ser Asn Lys Arg Glu Ile Pro Glu Val Leu Lys Asn
370 375 380
Ser Arg Ser Arg Pro Val Gly Thr Ser Met Phe Cys Phe Asp Gly Pro
385 390 395 400
Leu Thr Leu Val Ser Tyr Lys Pro Lys Pro Ala Lys Met Val Tyr Leu
405 410 415
Leu Ser Ser Cys Asp Glu Asp Ala Ser Ile Asn Glu Ser Thr Gly Lys
420 425 430
Pro Gln Met Val Met Tyr Tyr Asn Gln Thr Lys Gly Gly Val Asp Thr
435 440 445
Leu Asp Gln Met Cys Ser Val Met Thr Cys Ser Arg Lys Thr Asn Arg
450 455 460
Trp Pro Met Ala Leu Leu Tyr Gly Met Ile Asn Ile Ala Cys Ile Asn
465 470 475 480
Ser Phe Ile Ile Tyr Ser His Asn Val Ser Ser Lys Gly Glu Lys Val
485 490 495
Gln Ser Arg Lys Lys Phe Met Arg Asn Leu Tyr Met Gly Leu Thr Ser
500 505 510
Ser Phe Met Arg Lys Arg Leu Glu Ala Pro Thr Leu Lys Arg Tyr Leu
515 520 525
Arg Asp Asn Ile Ser Asn Ile Leu Pro Lys Glu Val Pro Gly Thr Ser
530 535 540
Asp Asp Ser Thr Glu Glu Pro Val Met Lys Lys Arg Thr Tyr Cys Ala
545 550 555 560
Tyr Cys Pro Ser Lys Ile Arg Arg Lys Ala Ser Ala Ser Cys Lys Lys
565 570 575
Cys Lys Lys Val Ile Cys Arg Glu His Asn Ile Asp Met Cys Gln Gly
580 585 590
Cys Phe
<210> 129
<211> 594
<212> PRT
<213> Artificial Sequence
<220>
<223> Regular modified 6 hyperactive PiggyBac aa sequence
<220>
<221> MISC_FEATURE
<222> (450)..(450)
<223> Wherein the amino acid can be Asp, Asn
<220>
<221> MISC_FEATURE
<222> (456)..(456)
<223> Wherein the amino acid can be Trp, Ala
<400> 129
Met Gly Ser Ser Leu Asp Asp Glu His Ile Leu Ser Ala Leu Leu Gln
1 5 10 15
Ser Asp Asp Glu Leu Val Gly Glu Asp Ser Asp Ser Glu Val Ser Asp
20 25 30
His Val Ser Glu Asp Asp Val Gln Ser Asp Thr Glu Glu Ala Phe Ile
35 40 45
Asp Glu Val His Glu Val Gln Pro Thr Ser Ser Gly Ser Glu Ile Leu
50 55 60
Asp Glu Gln Asn Val Ile Glu Gln Pro Gly Ser Ser Leu Ala Ser Asn
65 70 75 80
Arg Ile Leu Thr Leu Pro Gln Arg Thr Ile Arg Gly Lys Asn Lys His
85 90 95
Cys Trp Ser Thr Ser Lys Pro Thr Arg Arg Ser Arg Val Ser Ala Leu
100 105 110
Asn Ile Val Arg Ser Gln Arg Gly Pro Thr Arg Met Cys Arg Asn Ile
115 120 125
Tyr Asp Pro Leu Leu Cys Phe Lys Leu Phe Phe Thr Asp Glu Ile Ile
130 135 140
Ser Glu Ile Val Lys Trp Thr Asn Ala Glu Ile Ser Leu Lys Arg Arg
145 150 155 160
Glu Ser Met Thr Ser Ala Thr Phe Arg Asp Thr Asn Glu Asp Glu Ile
165 170 175
Tyr Ala Phe Phe Gly Ile Leu Val Met Thr Ala Val Arg Lys Asp Asn
180 185 190
His Met Ser Thr Asp Asp Leu Phe Asp Arg Ser Leu Ser Met Val Tyr
195 200 205
Val Ser Val Met Ser Arg Asp Arg Phe Asp Phe Leu Ile Arg Cys Leu
210 215 220
Arg Met Asp Asp Lys Ser Ile Arg Pro Thr Leu Arg Glu Asn Asp Val
225 230 235 240
Phe Thr Pro Val Arg Lys Ile Trp Asp Leu Phe Ile His Gln Cys Ile
245 250 255
Gln Asn Tyr Thr Pro Gly Ala His Leu Thr Ile Asp Glu Gln Leu Leu
260 265 270
Gly Phe Ala Gly Arg Cys Pro Phe Arg Val Tyr Ile Pro Asn Lys Pro
275 280 285
Ser Lys Tyr Gly Ile Lys Ile Leu Met Met Cys Asp Ser Gly Thr Lys
290 295 300
Tyr Met Ile Asn Gly Met Pro Tyr Leu Gly Arg Gly Thr Gln Thr Asn
305 310 315 320
Gly Val Pro Leu Gly Glu Tyr Tyr Val Lys Glu Leu Ser Lys Pro Val
325 330 335
His Gly Ser Cys Arg Asn Ile Thr Cys Asp Asn Trp Phe Thr Ala Ile
340 345 350
Pro Leu Ala Lys Asn Leu Leu Gln Glu Pro Tyr Lys Leu Thr Ile Val
355 360 365
Gly Thr Val Ala Ser Asn Ala Arg Glu Ile Pro Glu Val Leu Lys Asn
370 375 380
Ser Arg Ser Arg Pro Val Gly Thr Ser Met Phe Cys Phe Asp Gly Pro
385 390 395 400
Leu Thr Leu Val Ser Tyr Lys Pro Lys Pro Ala Lys Met Val Tyr Leu
405 410 415
Leu Ser Ser Cys Asp Glu Asp Ala Ser Ile Asn Glu Ser Thr Gly Lys
420 425 430
Pro Gln Met Val Met Tyr Tyr Asn Gln Thr Lys Gly Gly Val Asp Thr
435 440 445
Leu Asp Gln Met Cys Ser Val Met Thr Cys Ser Arg Lys Thr Asn Arg
450 455 460
Trp Pro Met Ala Leu Leu Tyr Gly Met Ile Asn Ile Ala Cys Ile Asn
465 470 475 480
Ser Phe Ile Ile Tyr Ser His Asn Val Ser Ser Lys Gly Glu Lys Val
485 490 495
Gln Ser Arg Lys Lys Phe Met Arg Asn Leu Tyr Met Gly Leu Thr Ser
500 505 510
Ser Phe Met Arg Lys Arg Leu Glu Ala Pro Thr Leu Lys Arg Tyr Leu
515 520 525
Arg Asp Asn Ile Ser Asn Ile Leu Pro Lys Glu Val Pro Gly Thr Ser
530 535 540
Asp Asp Ser Thr Glu Glu Pro Val Met Lys Lys Arg Thr Tyr Cys Ala
545 550 555 560
Tyr Cys Pro Ser Lys Ile Arg Arg Lys Ala Ser Ala Ser Cys Lys Lys
565 570 575
Cys Lys Lys Val Ile Cys Arg Glu His Asn Ile Asp Met Cys Gln Gly
580 585 590
Cys Phe
<210> 130
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> Linker aa sequence
<400> 130
Lys Leu Ala Gly Gly Ala Pro Ala Val Gly Gly Gly Pro Lys
1 5 10
<210> 131
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<223> Linker aa sequence
<400> 131
Glu Phe Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
1 5 10 15
Ser Gln Phe
<210> 132
<211> 20
<212> PRT
<213> Artificial Sequence
<220>
<223> Primer SV40pA-R
<400> 132
Gly Ala Ala Ala Thr Thr Thr Gly Thr Gly Ala Thr Gly Cys Thr Ala
1 5 10 15
Thr Thr Gly Cys
20
<210> 133
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> Linker
<220>
<221> MISC_FEATURE
<222> (1)..(5)
<223> Wherein the sequence can be repeated 1 to 50 times
<400> 133
Gly Gly Gly Gly Ser
1 5
<210> 134
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> Linker
<220>
<221> MISC_FEATURE
<222> (1)..(5)
<223> Wherein the sequence can be repeated 1 to 50 times
<400> 134
Glu Ala Ala Ala Lys
1 5
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| ИНДУЦИРУЕМЫЕ КАСПАЗЫ И СПОСОБЫ ИСПОЛЬЗОВАНИЯ | 2017 |
|
RU2757058C2 |
| ИНТЕГРАЦИЯ КОНСТРУКЦИЙ НУКЛЕИНОВОЙ КИСЛОТЫ В ЭУКАРИОТИЧЕСКИЕ КЛЕТКИ С ТРАНСПОЗАЗОЙ ИЗ ORYZIAS | 2020 |
|
RU2817770C2 |
| ТРАНСПОЗИЦИЯ КОНСТРУКЦИЙ НУКЛЕИНОВОЙ КИСЛОТЫ В ЭУКАРИОТИЧЕСКИЕ ГЕНОМЫ С ТРАНСПОЗАЗОЙ ИЗ AMYELOIS | 2020 |
|
RU2814721C2 |
| УЛУЧШЕННЫЕ ЭКСПРЕССИЯ И ПРОЦЕССИНГ ТРАНСГЕНА | 2014 |
|
RU2711505C2 |
| КЛЕТКИ ДЛЯ ИММУНОТЕРАПИИ, СКОНСТРУИРОВАННЫЕ ДЛЯ НАЦЕЛИВАНИЯ НА АНТИГЕН, ПРИСУТСТВУЮЩИЙ ОДНОВРЕМЕННО НА ИММУННЫХ КЛЕТКАХ И НА ПАТОЛОГИЧЕСКИХ КЛЕТКАХ | 2015 |
|
RU2714258C2 |
| HPV-СПЕЦИФИЧЕСКИЕ СВЯЗЫВАЮЩИЕ МОЛЕКУЛЫ | 2018 |
|
RU2804664C2 |
| СПЕЦИФИЧНЫЕ К MUC16 ХИМЕРНЫЕ АНТИГЕННЫЕ РЕЦЕПТОРЫ И ИХ ПРИМЕНЕНИЯ | 2019 |
|
RU2795198C2 |
| КОМПОЗИЦИИ И СПОСОБЫ РЕДАКТИРОВАНИЯ ГЕНОВ | 2019 |
|
RU2804665C2 |
| НОВЫЕ ФЕРМЕНТЫ И СИСТЕМЫ CRISPR | 2016 |
|
RU2737537C2 |
| НОВЫЕ ФЕРМЕНТЫ И СИСТЕМЫ CRISPR | 2023 |
|
RU2832308C2 |
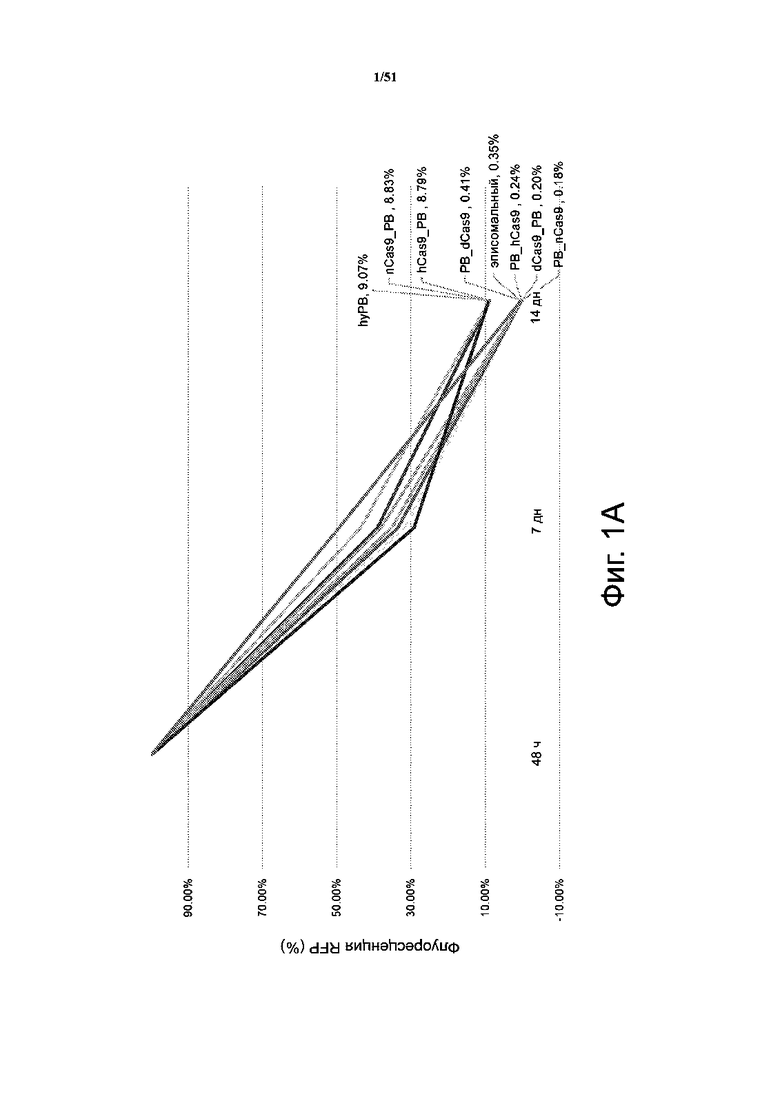
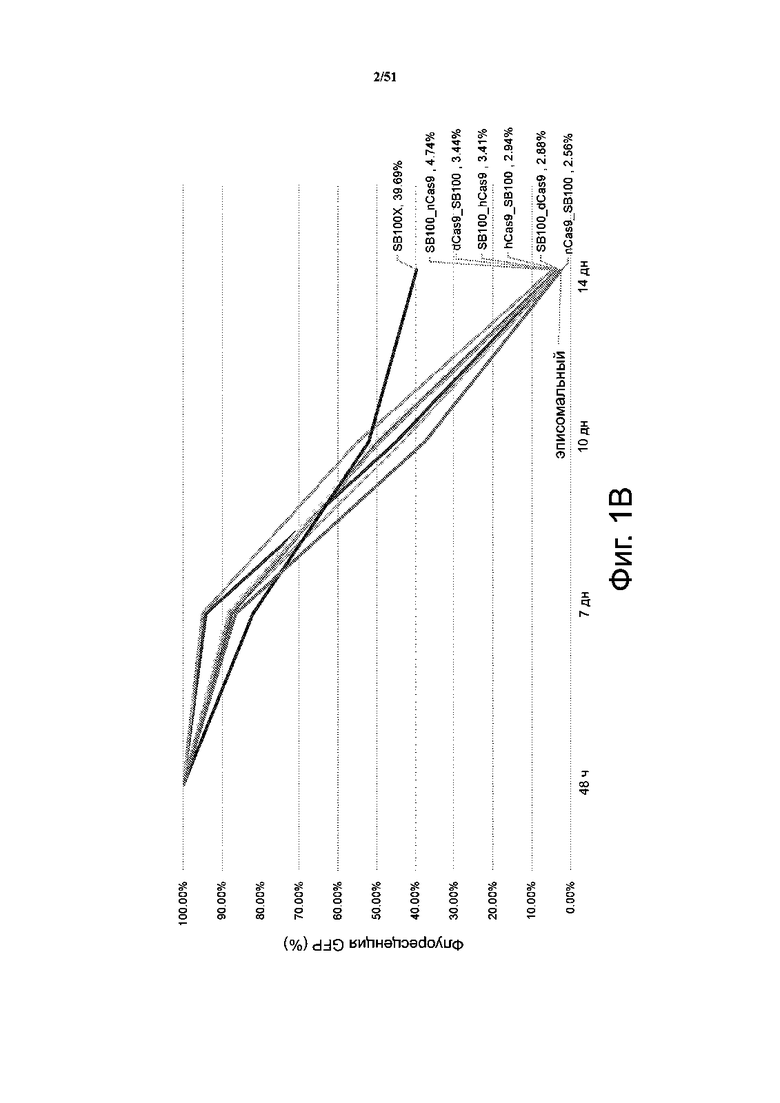
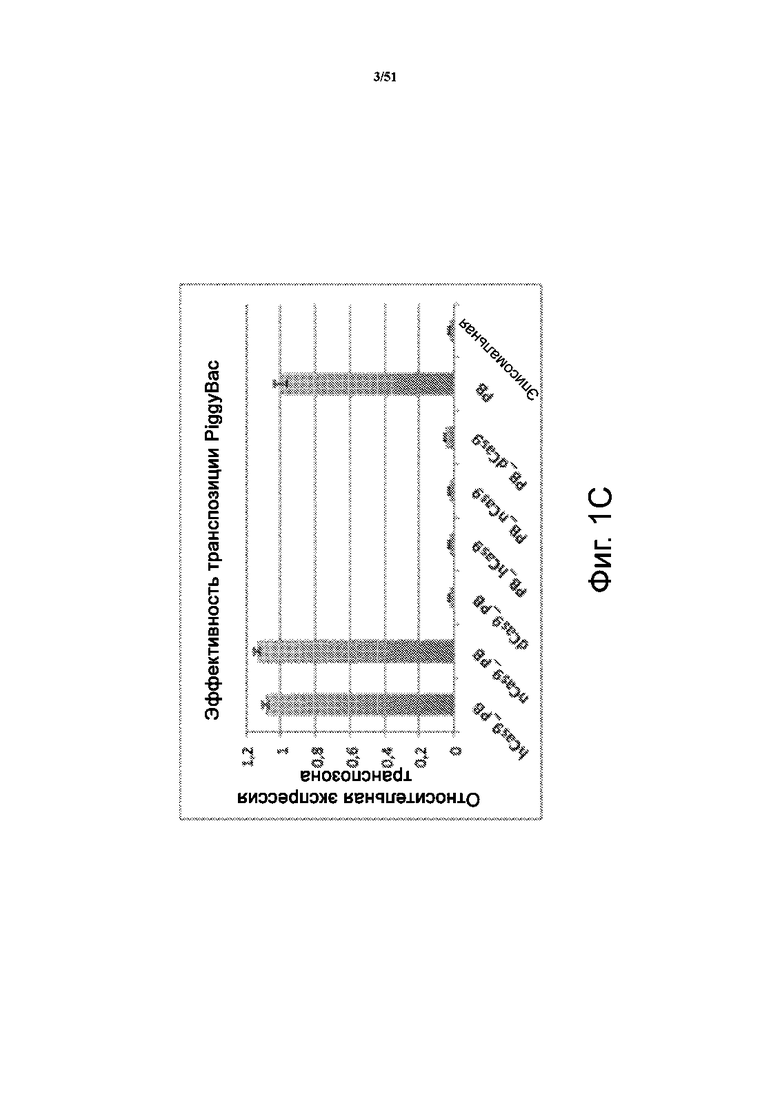
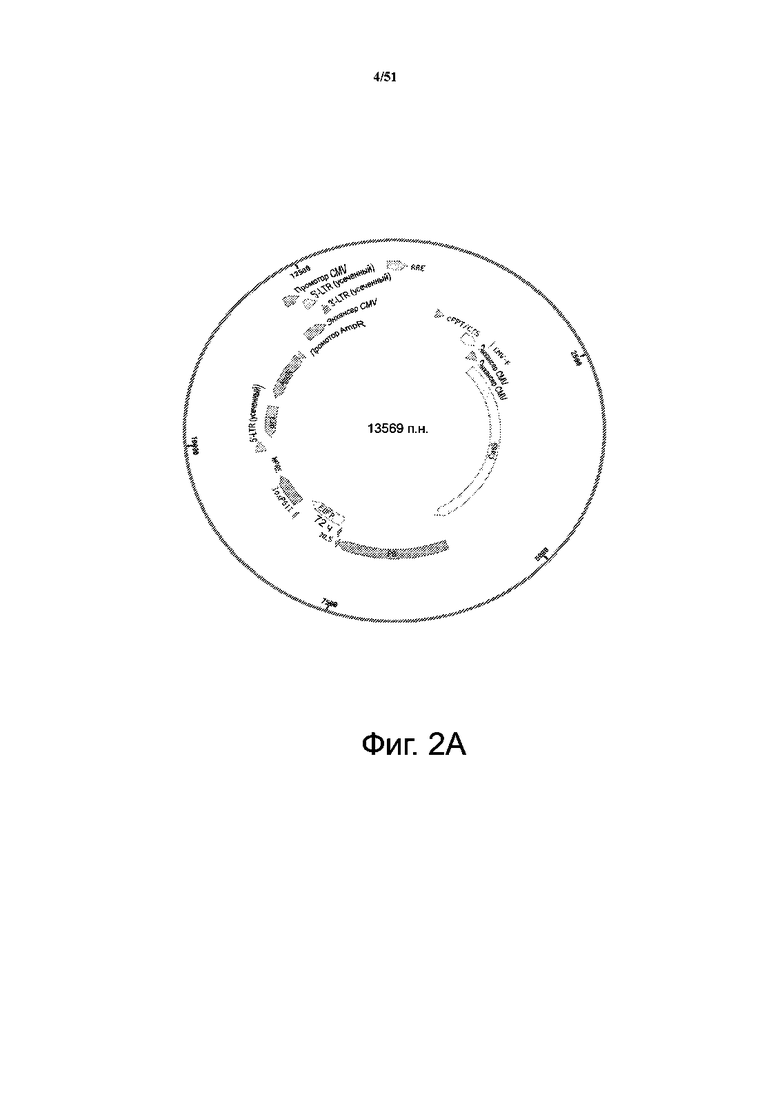
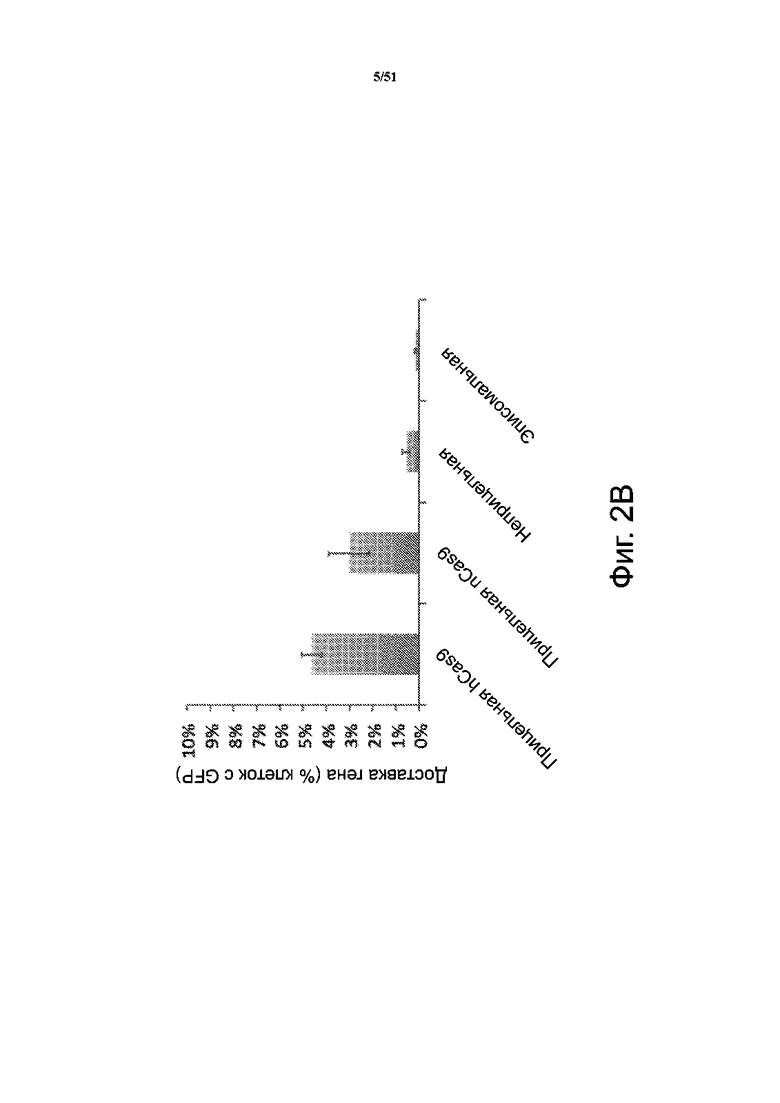
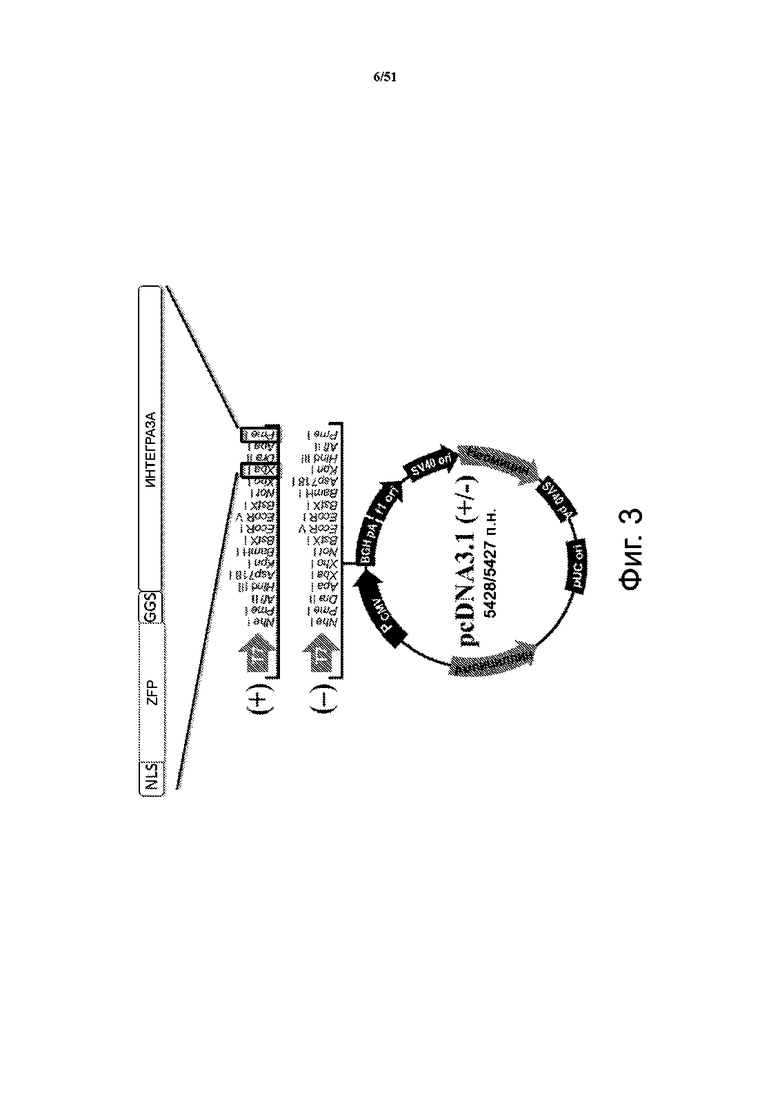
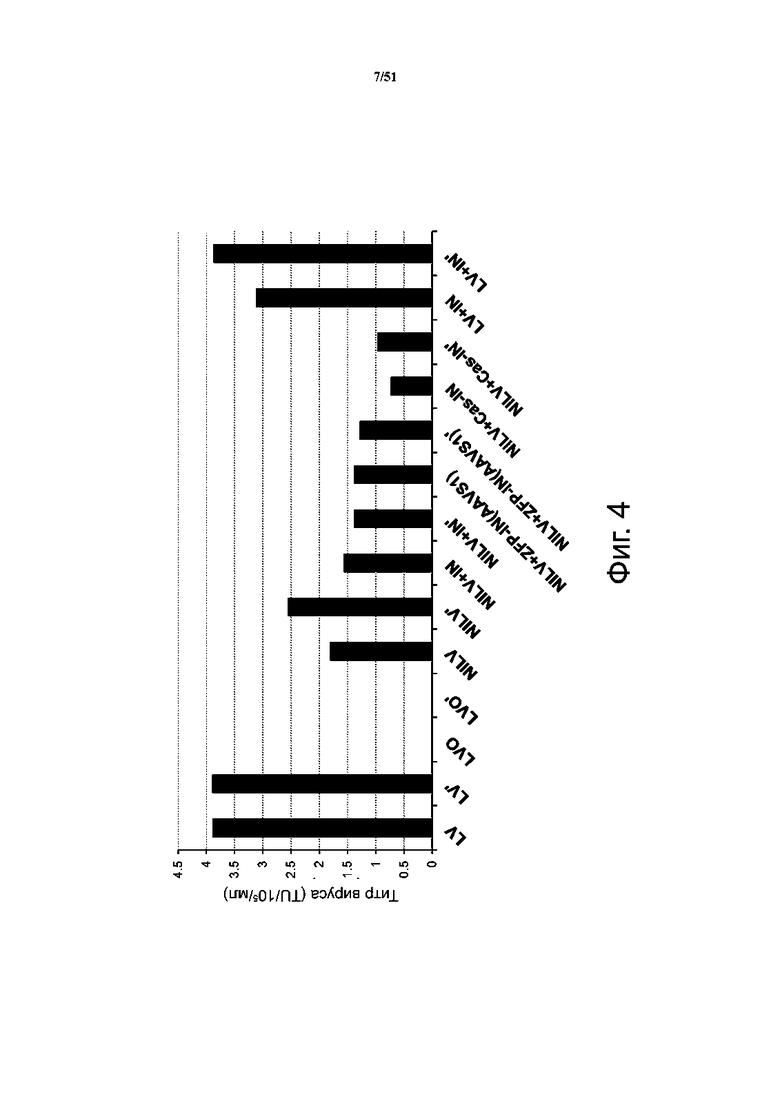
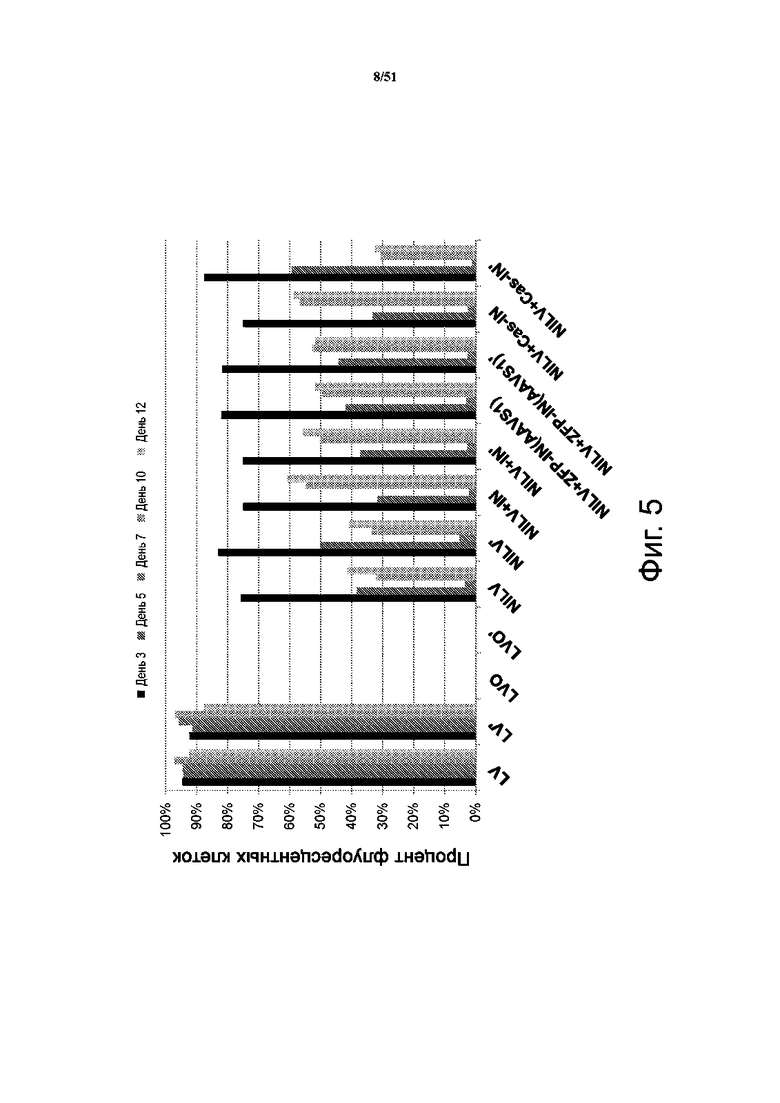

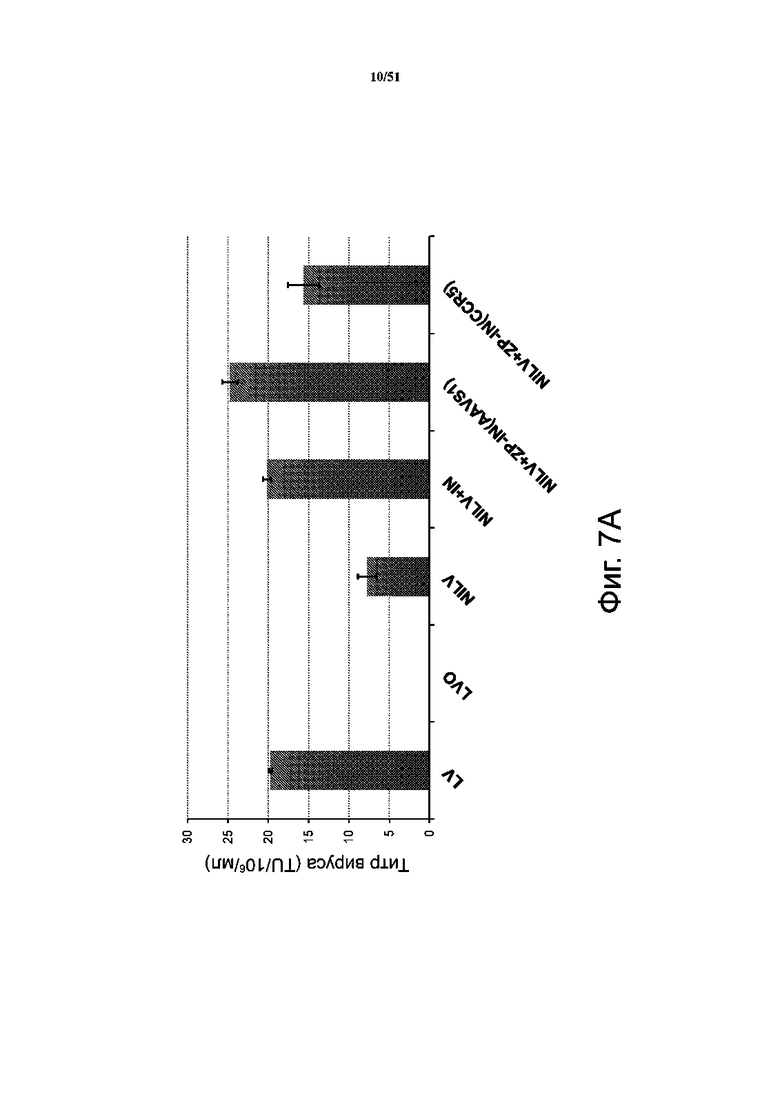
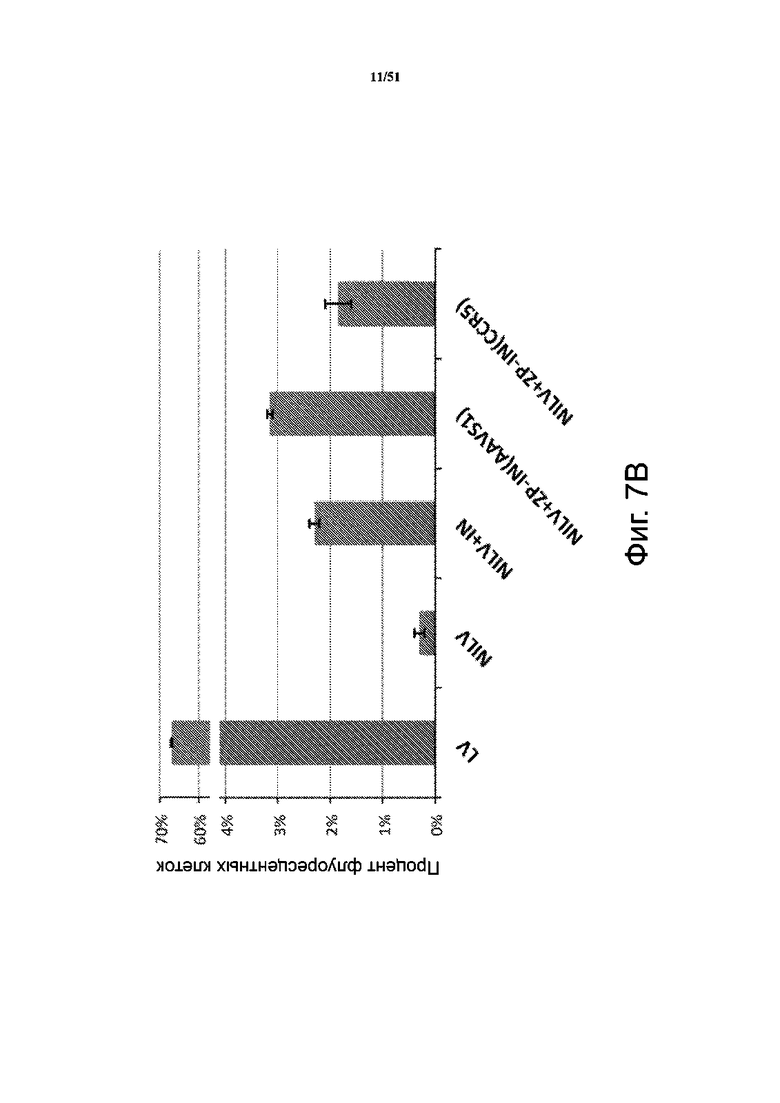
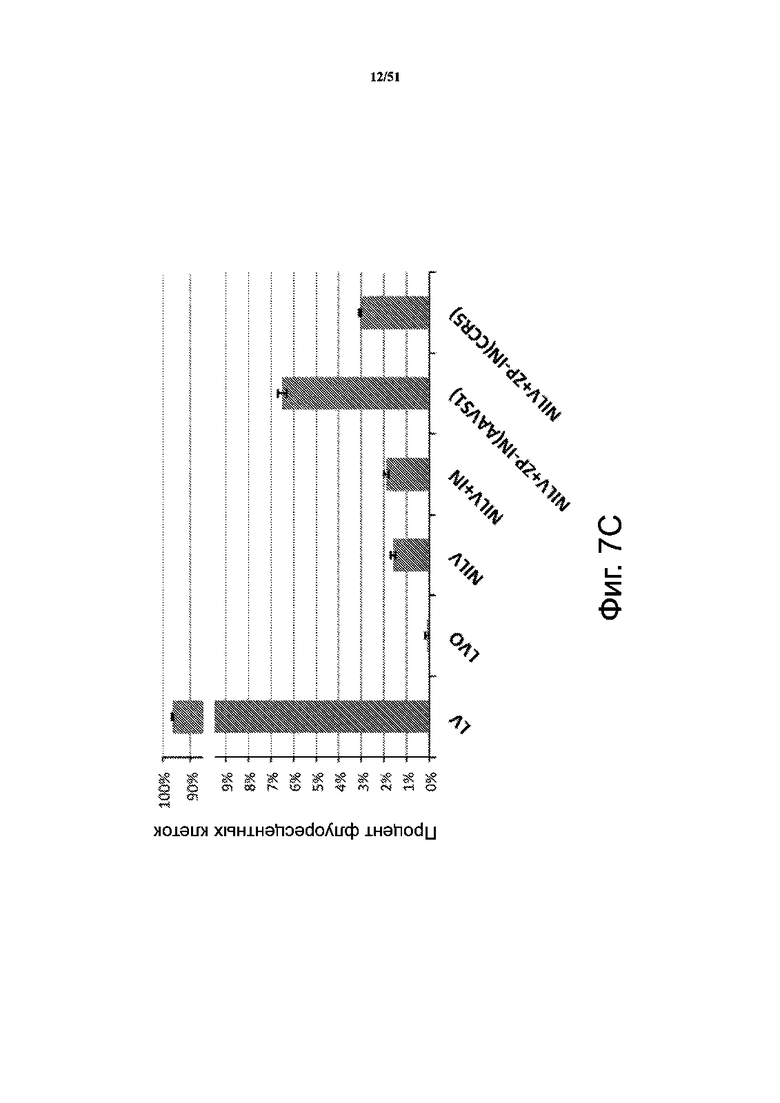
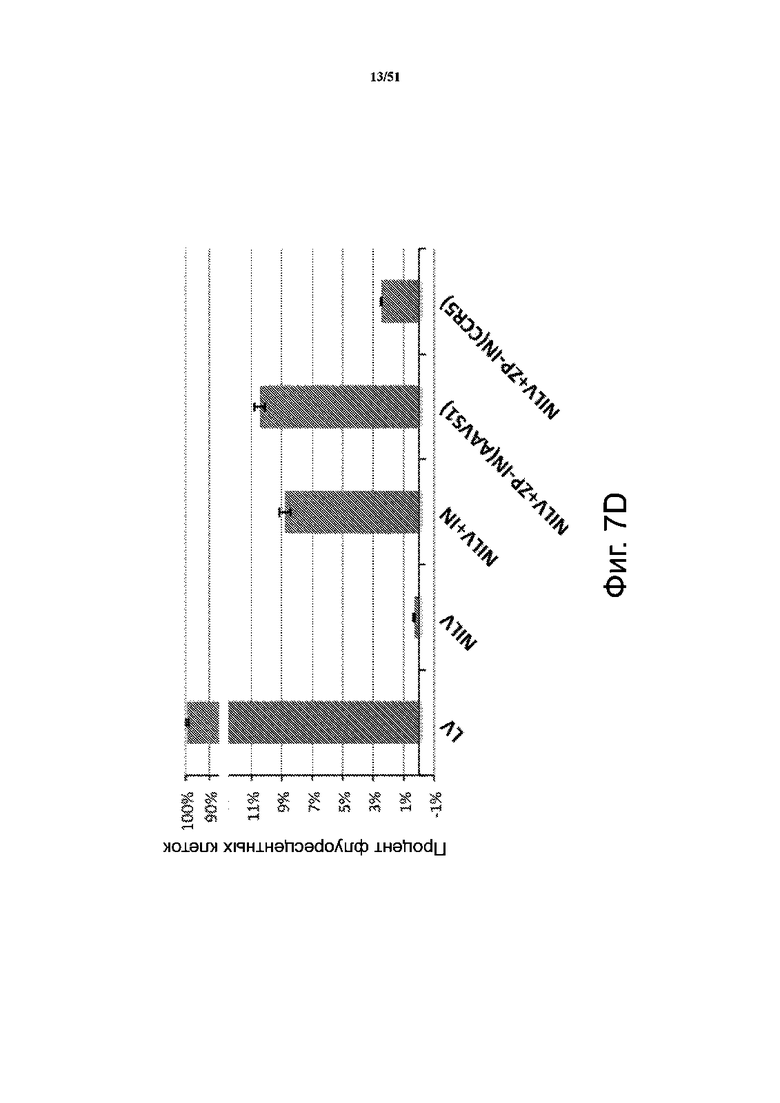
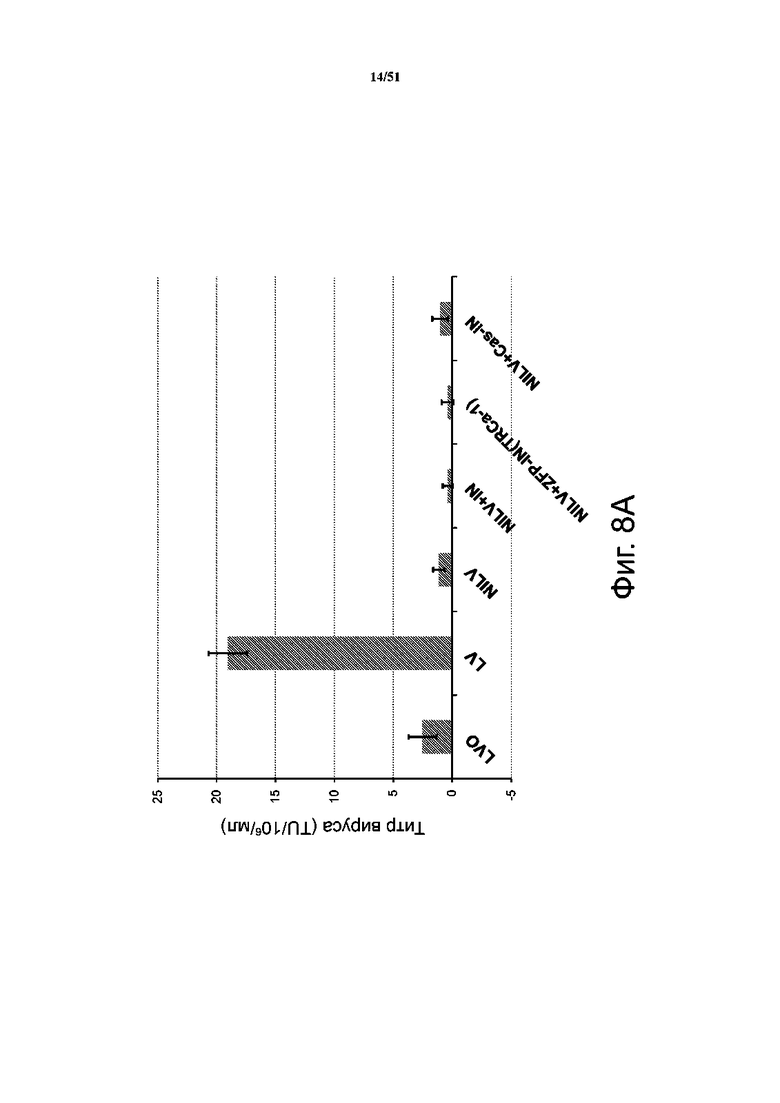
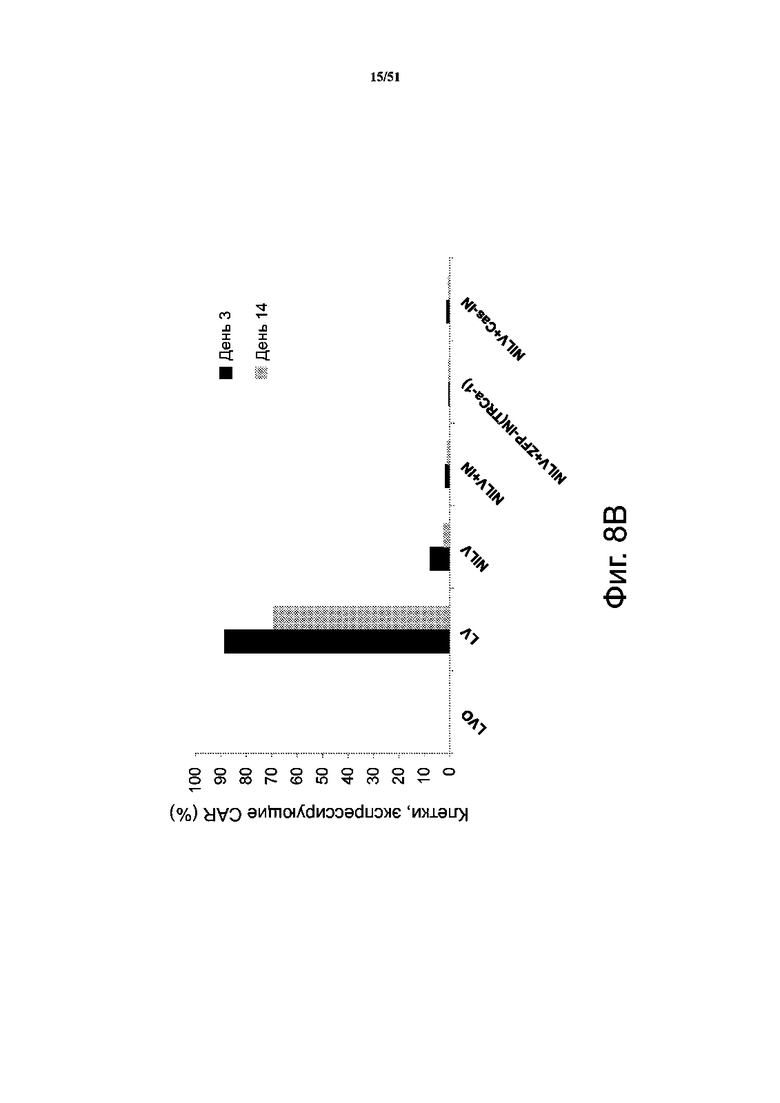
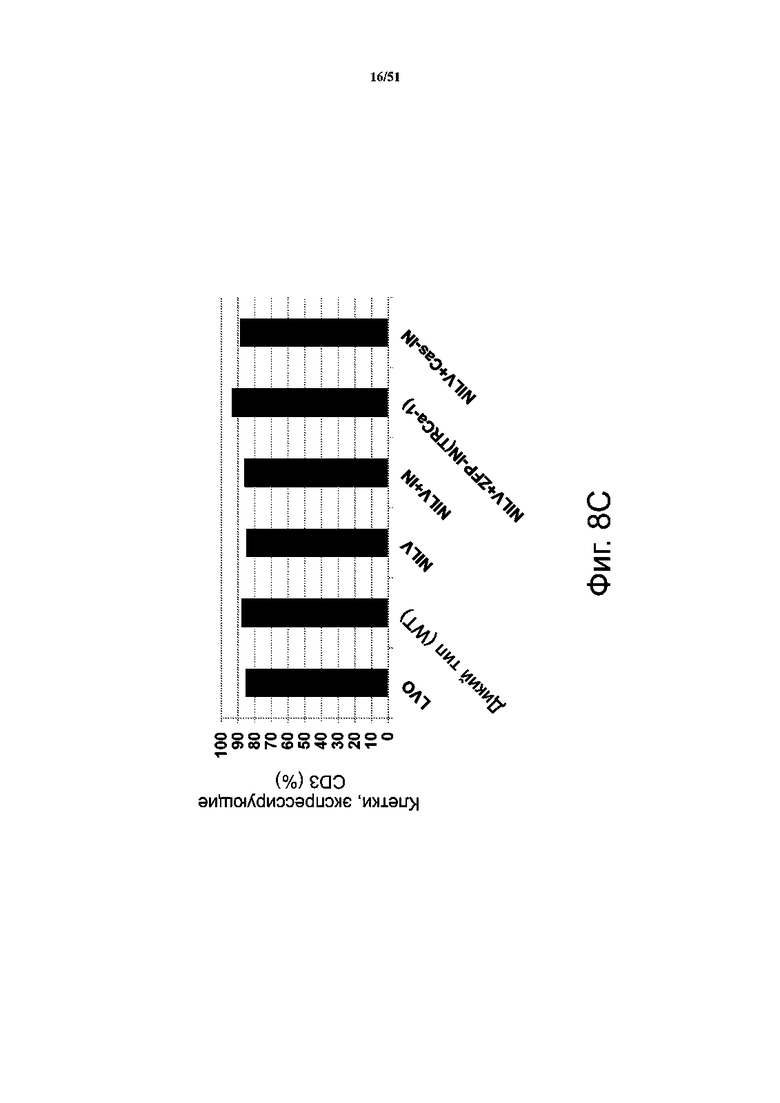
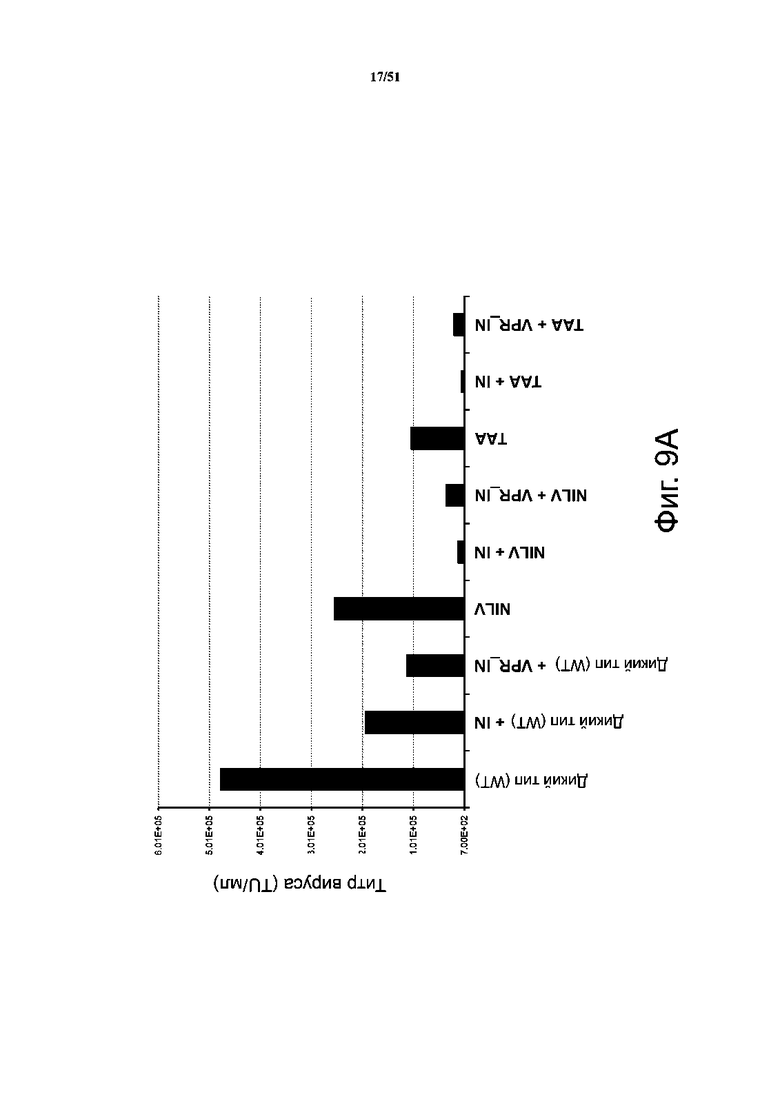
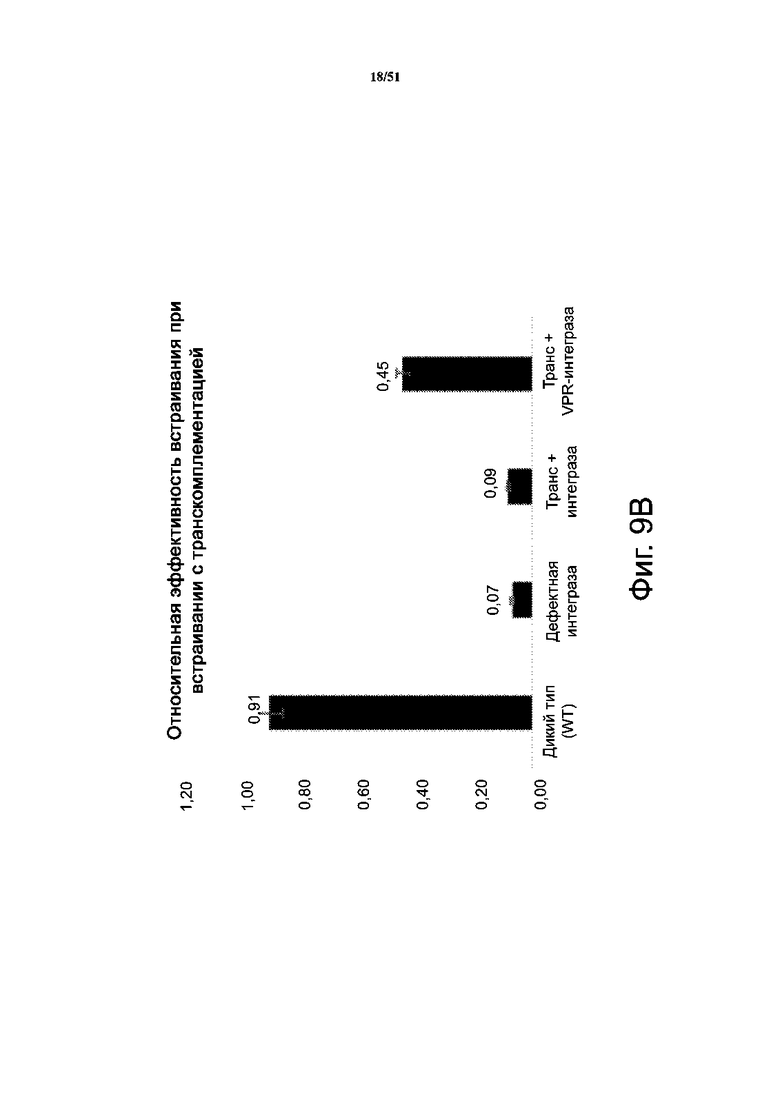
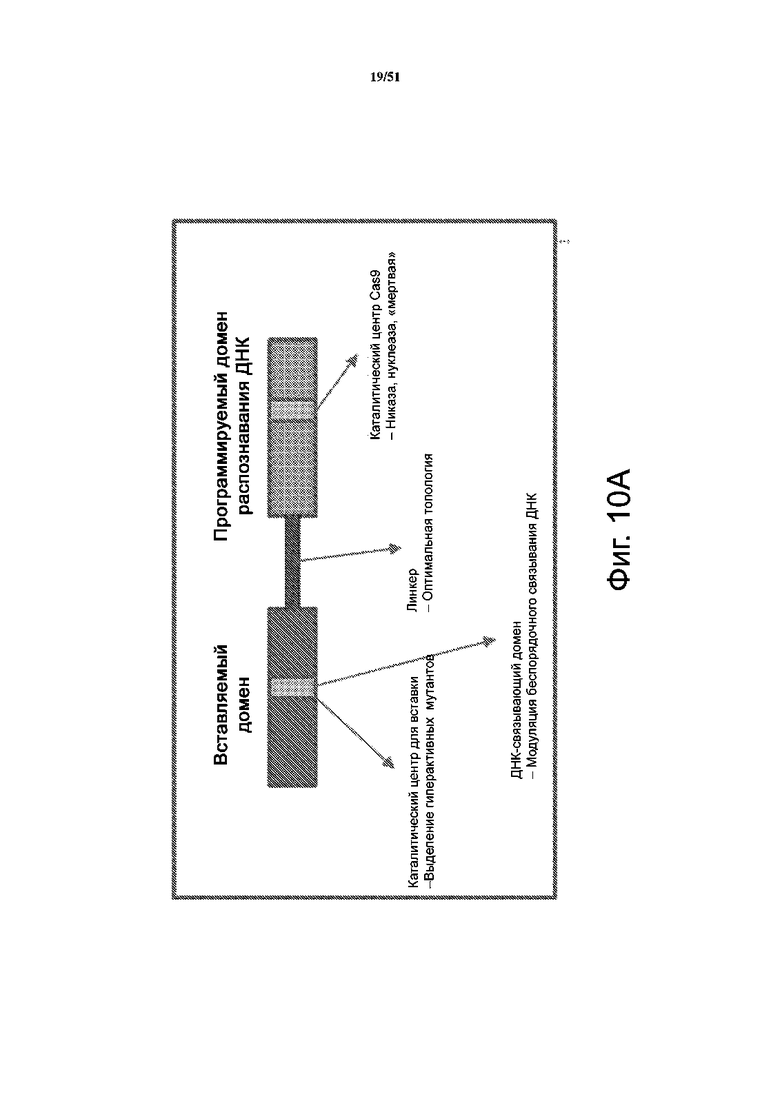
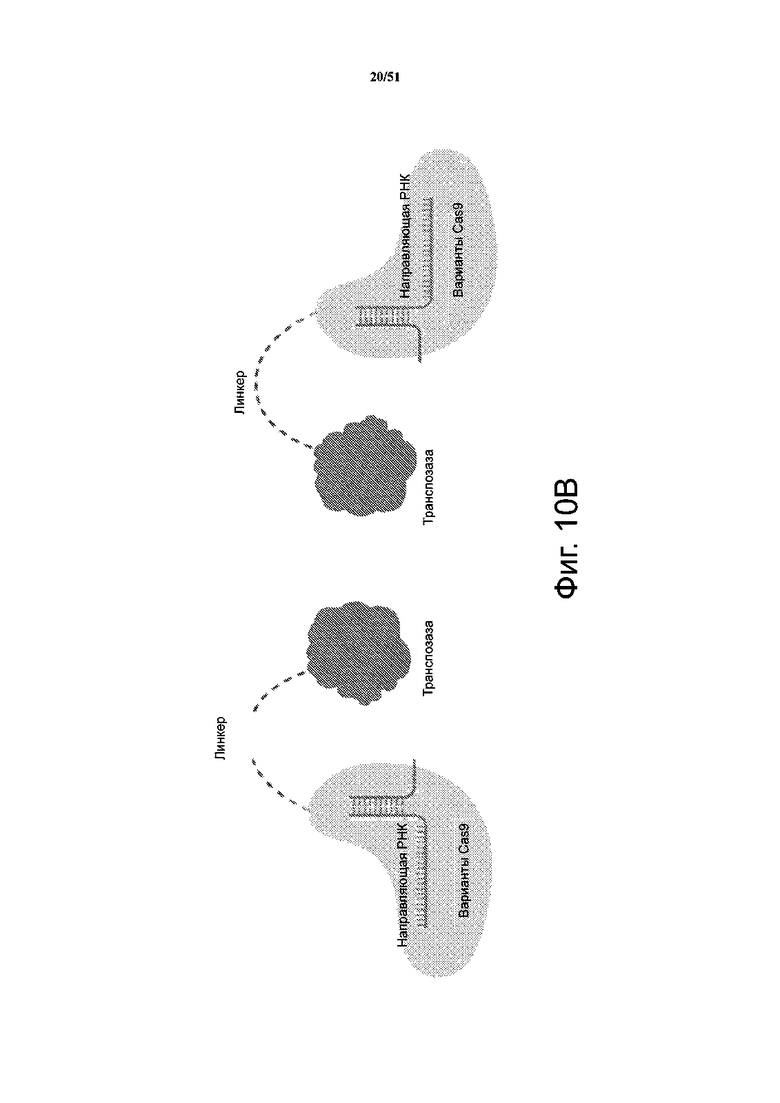
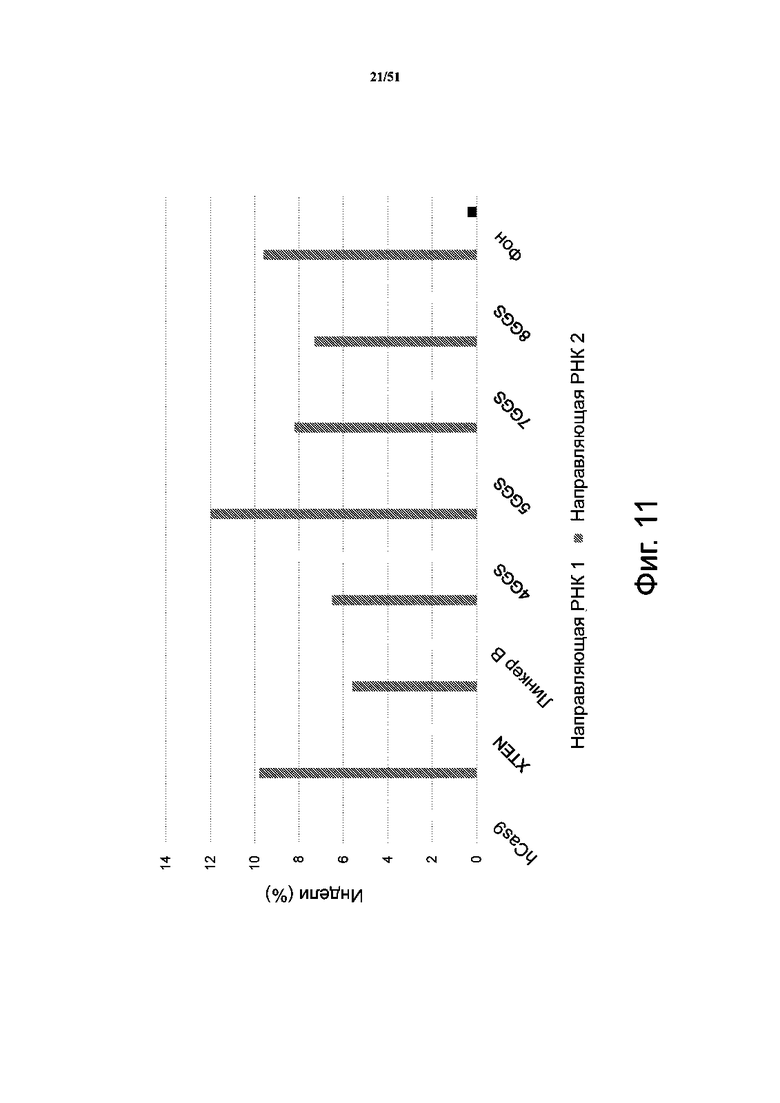
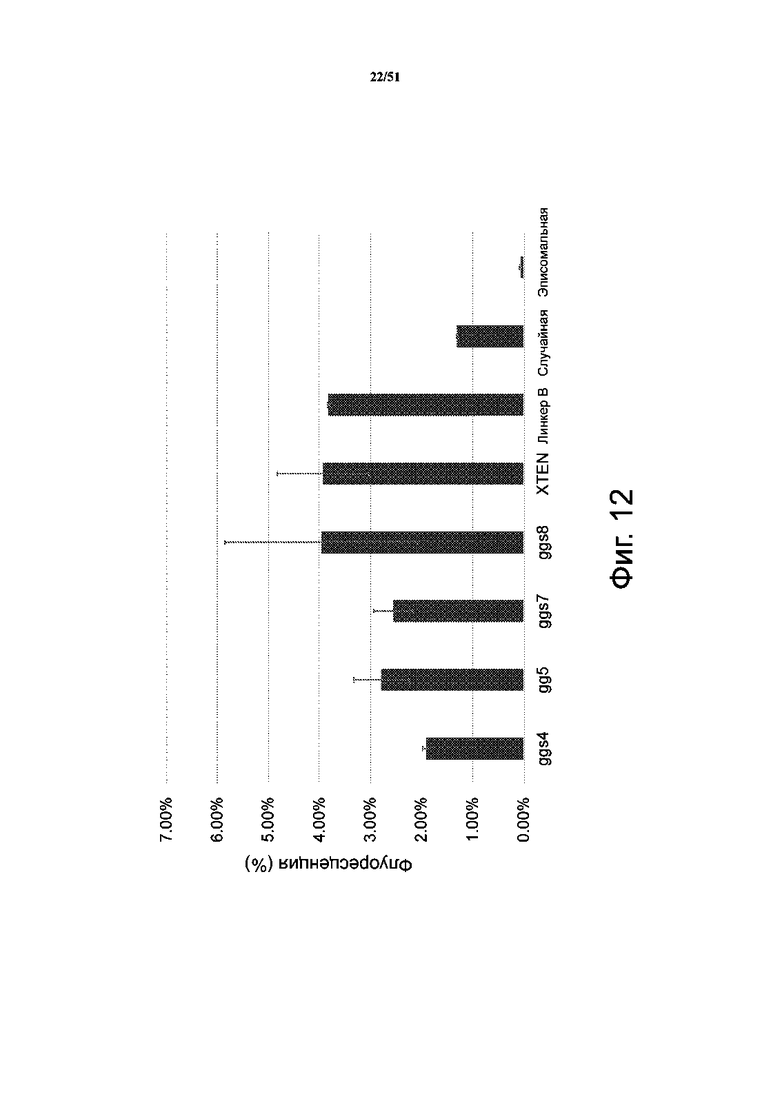
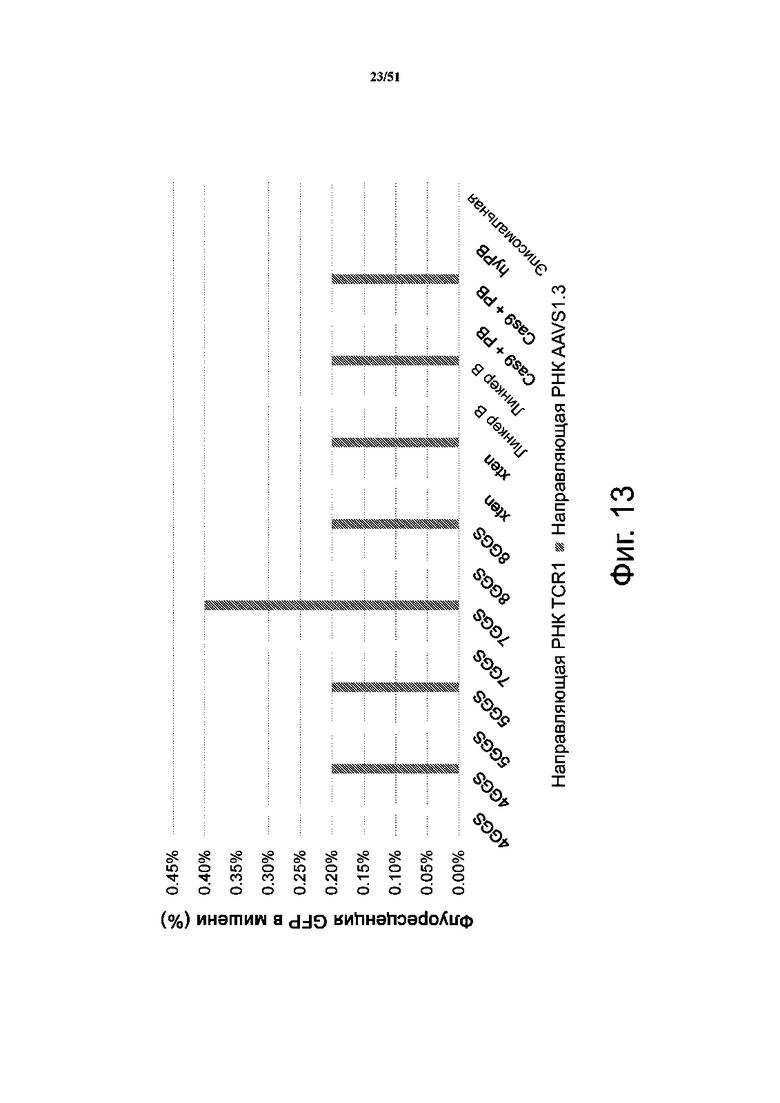
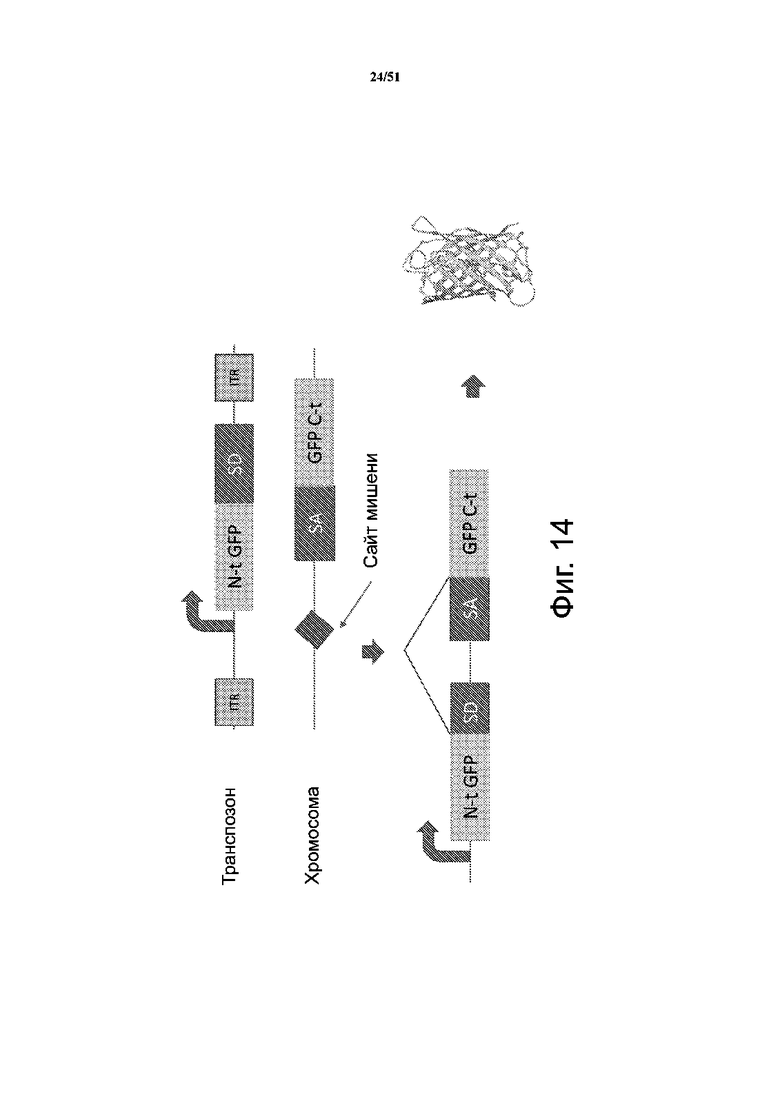
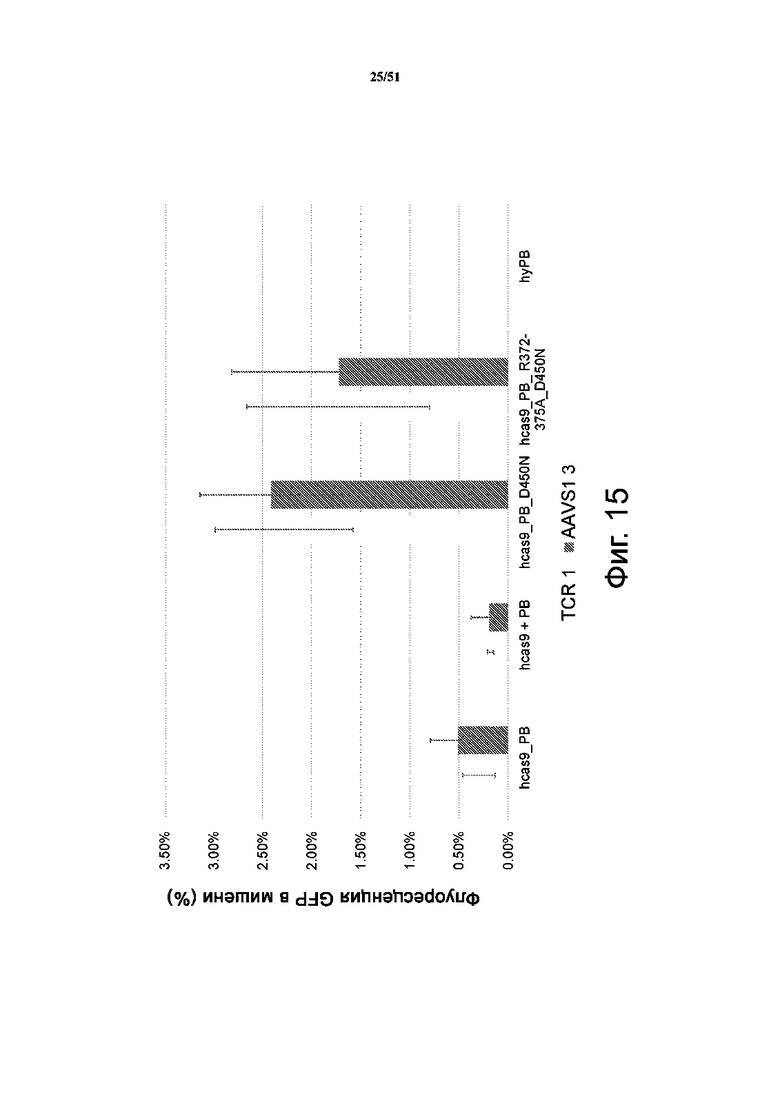
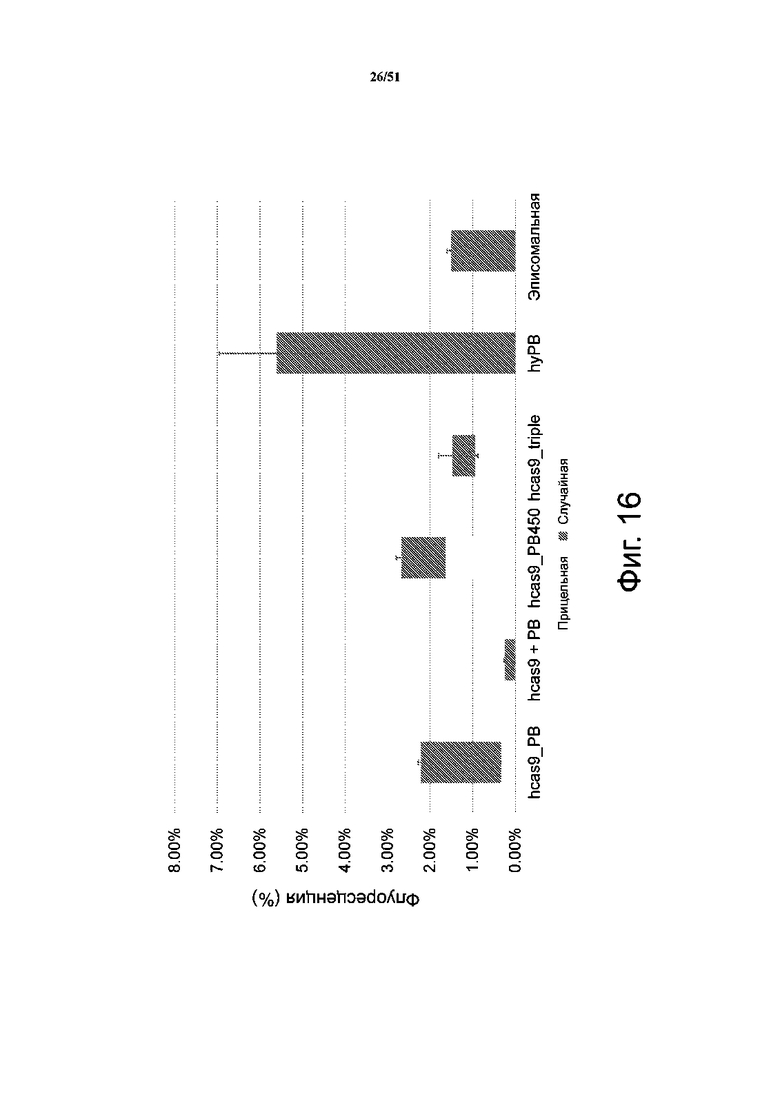
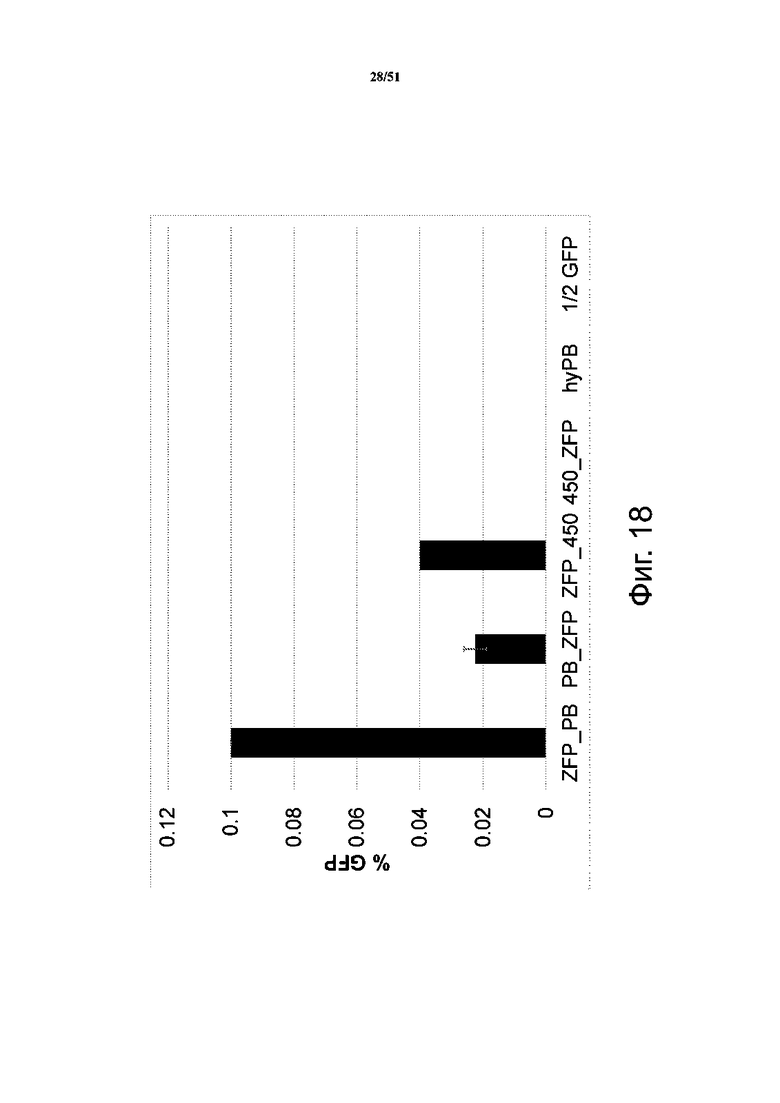
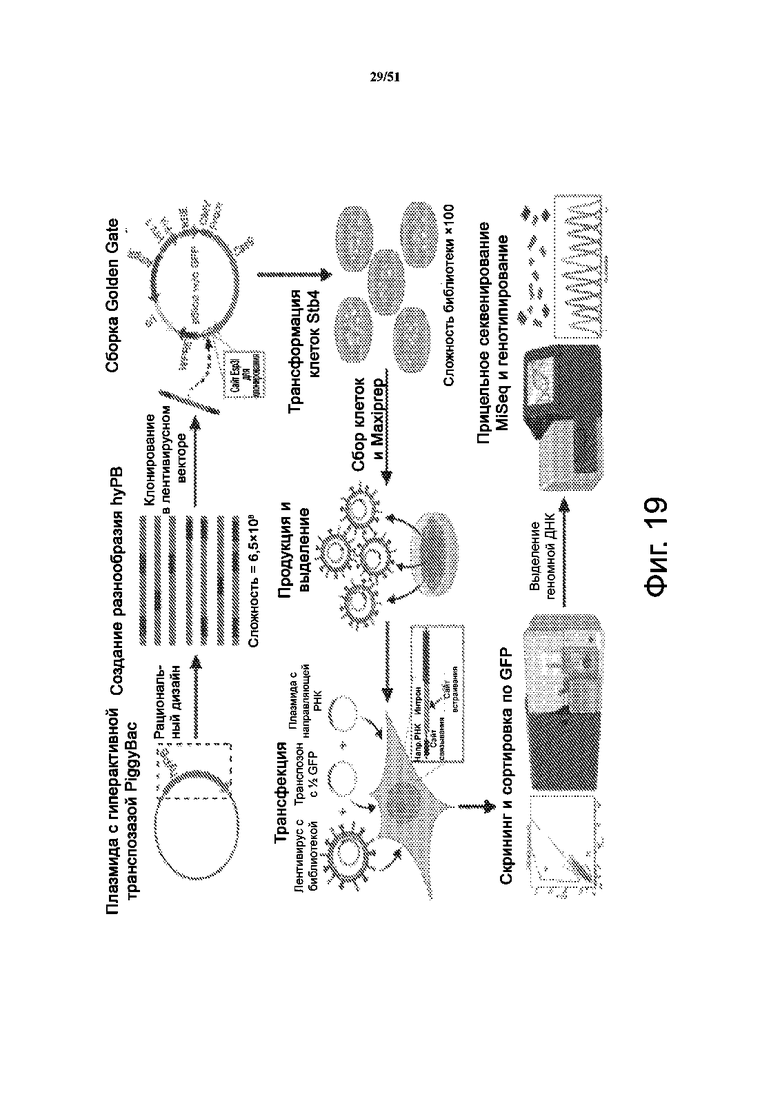

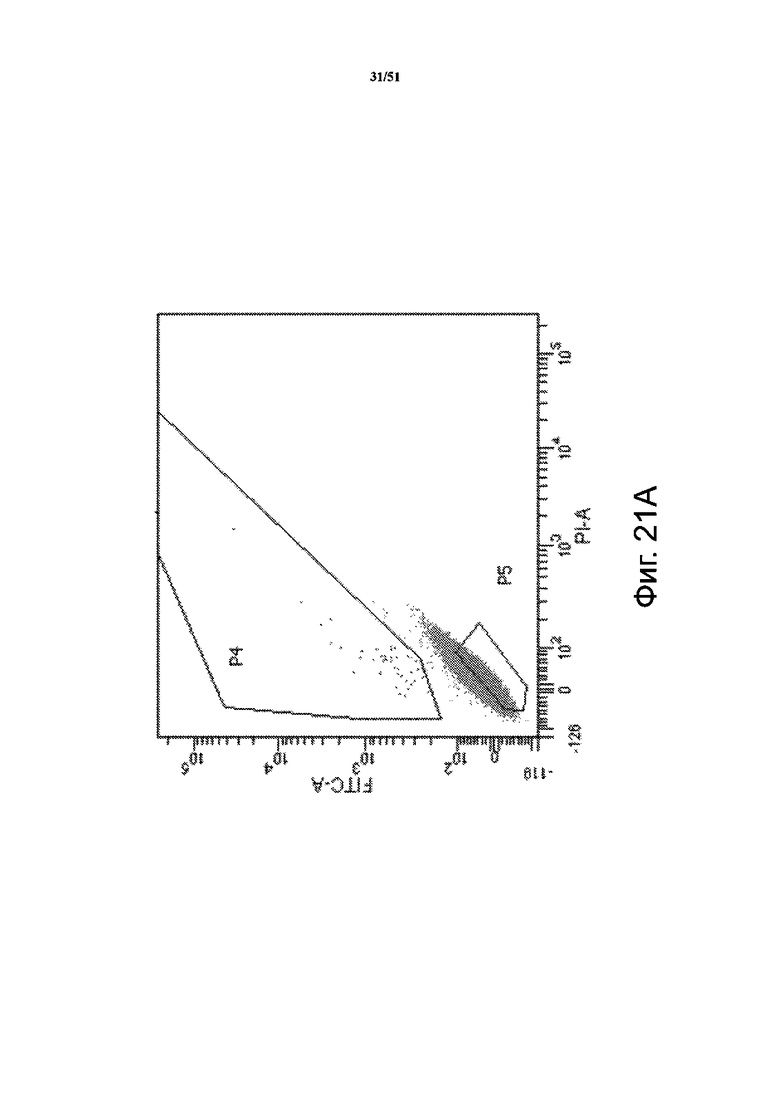
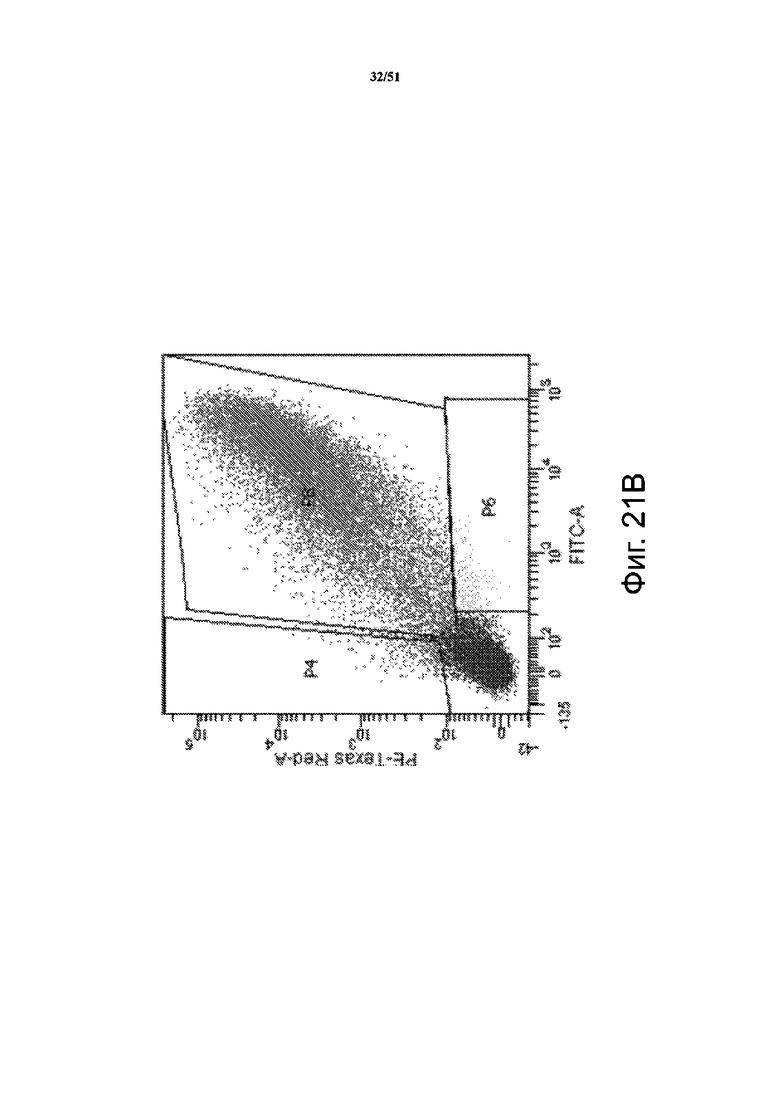


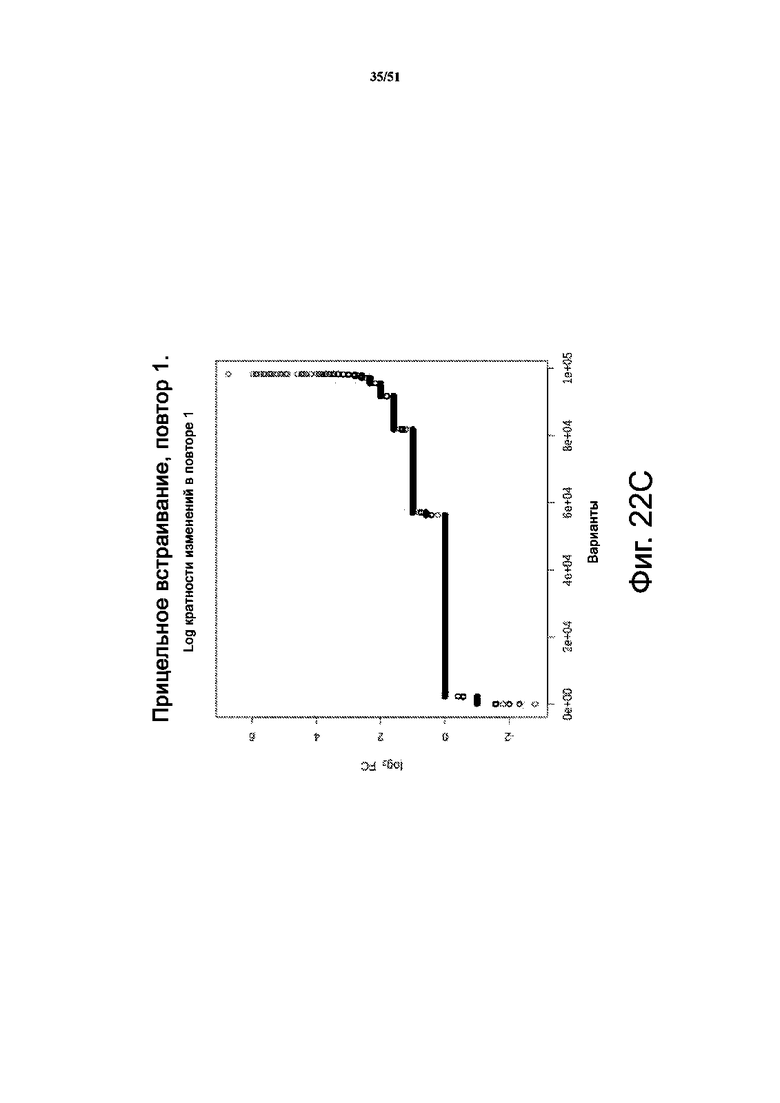
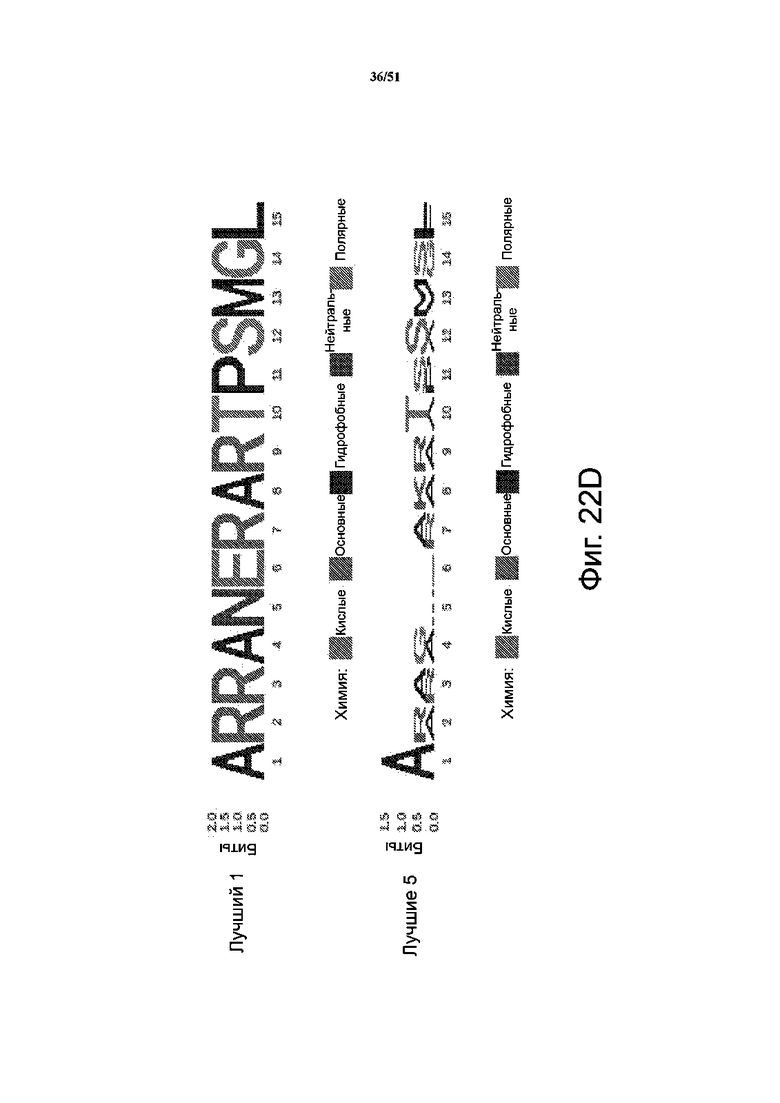
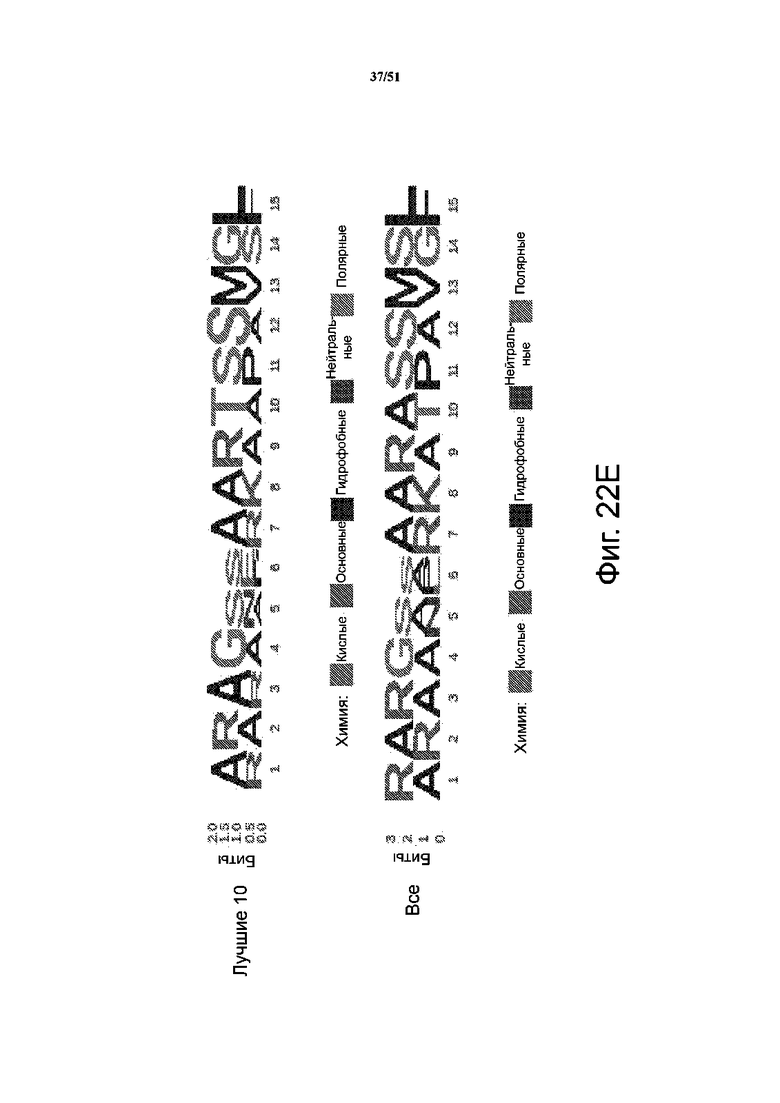
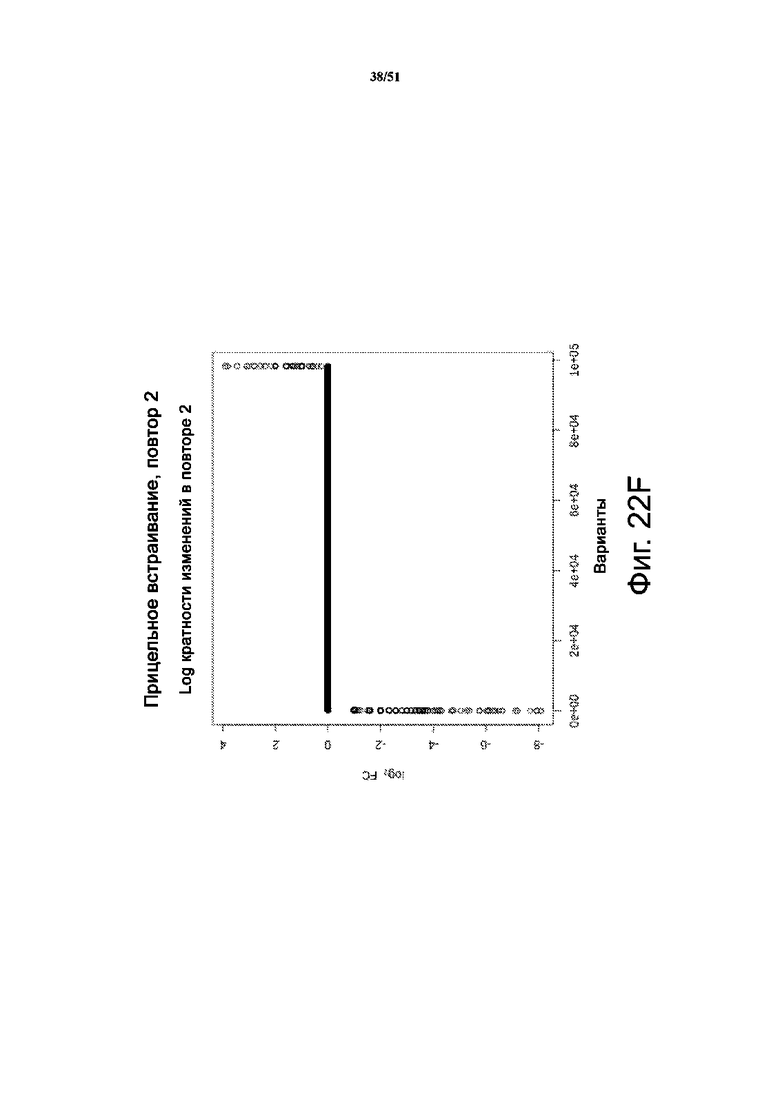
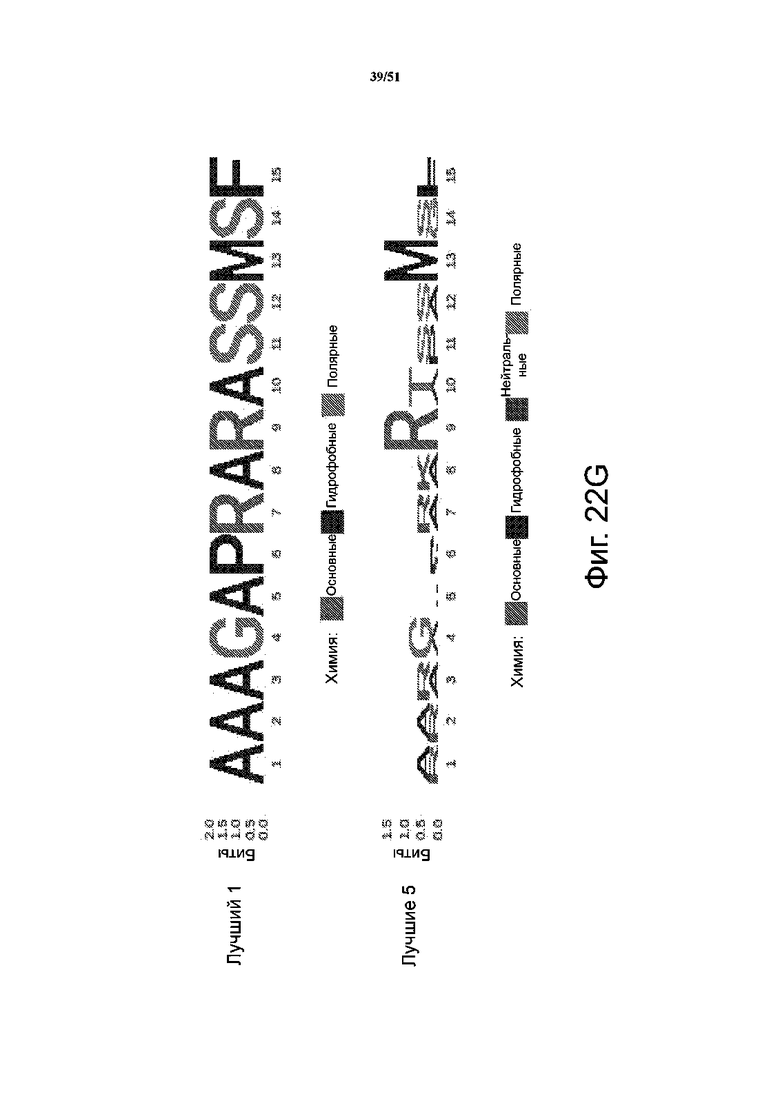
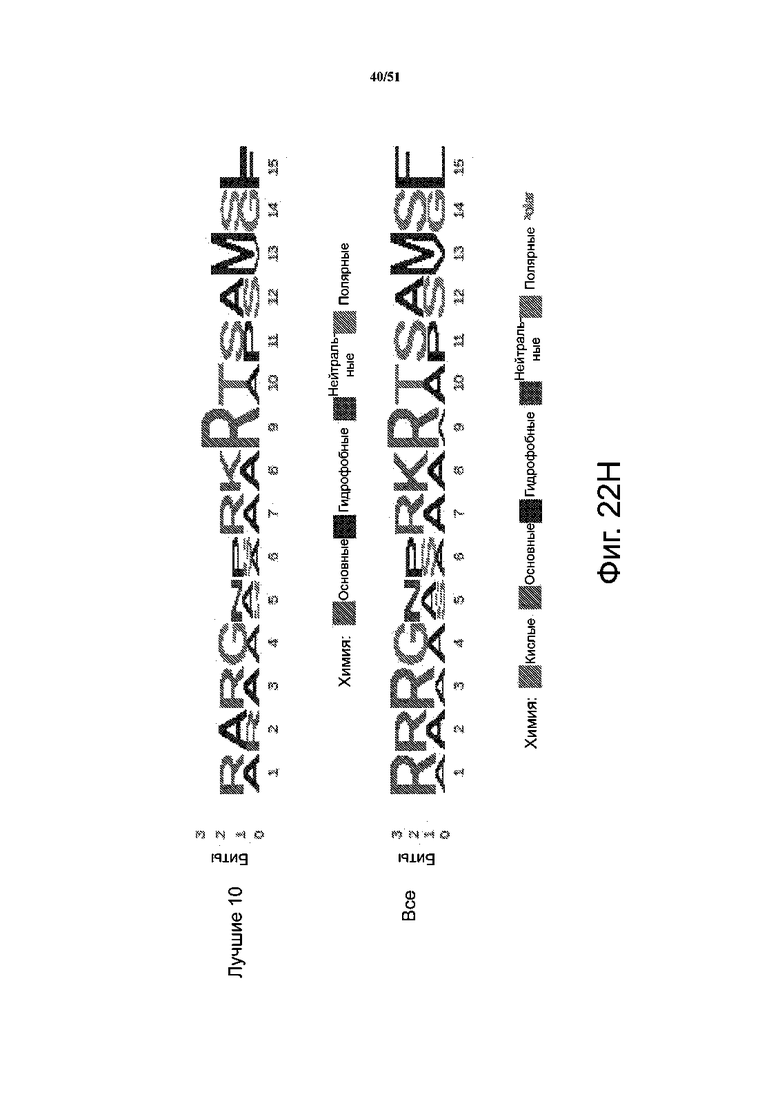
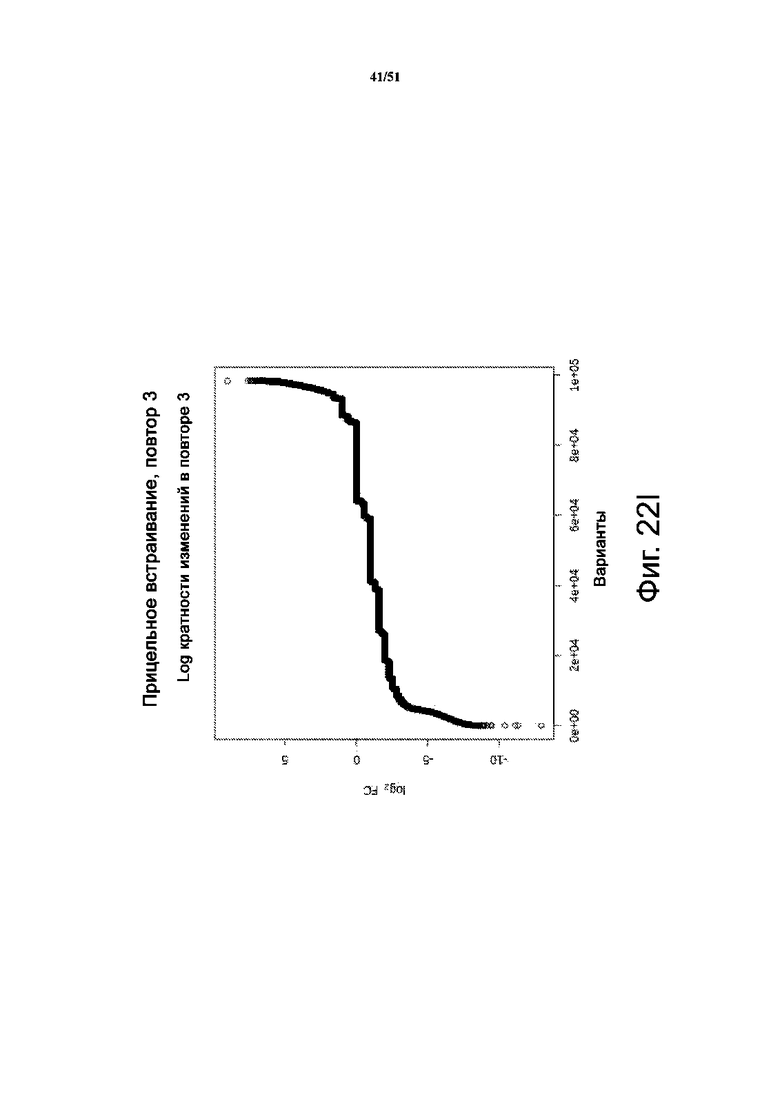
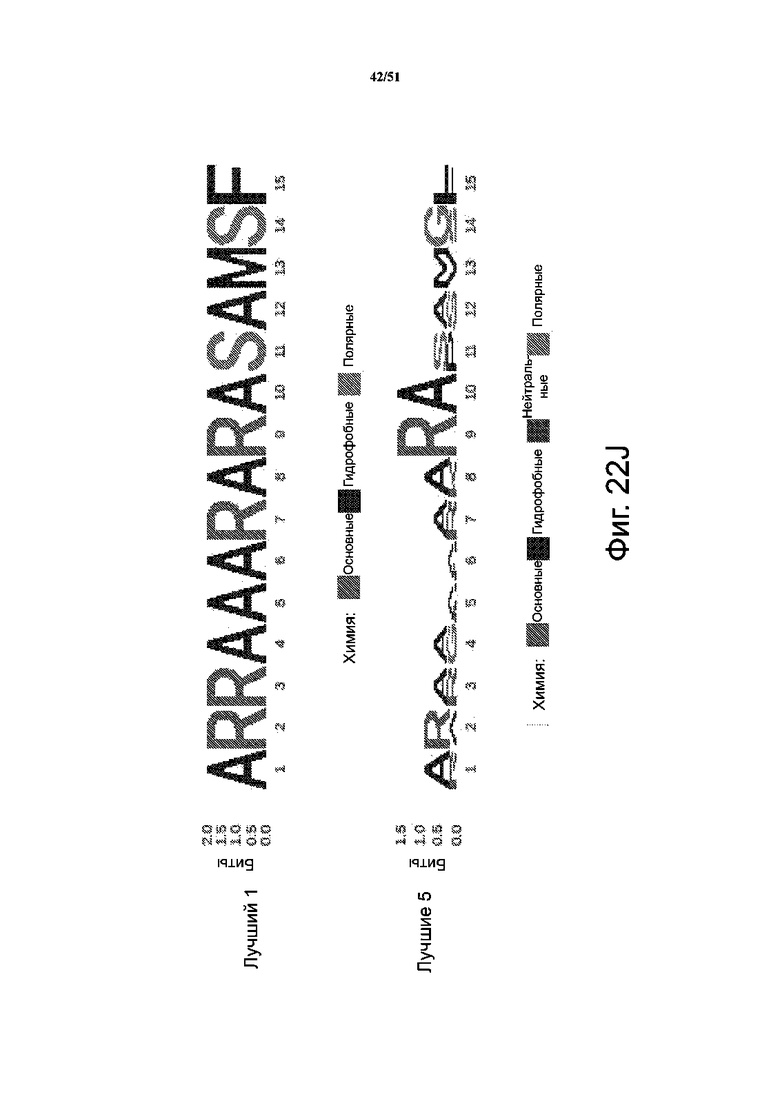
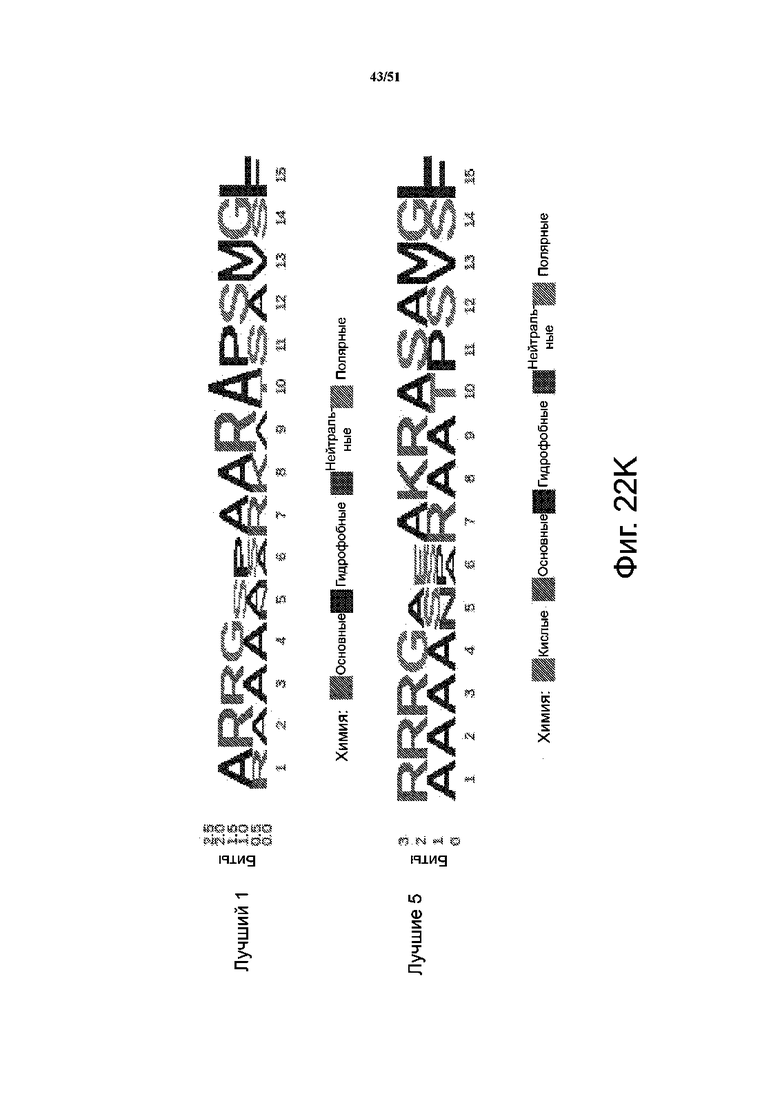


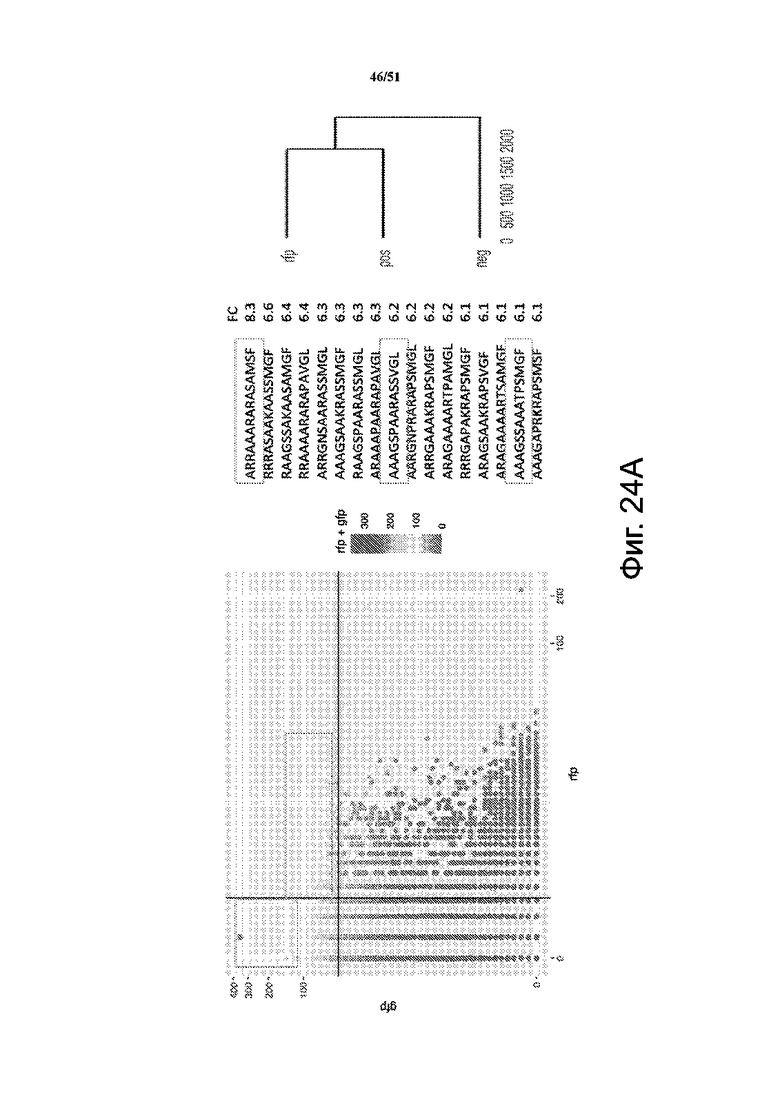


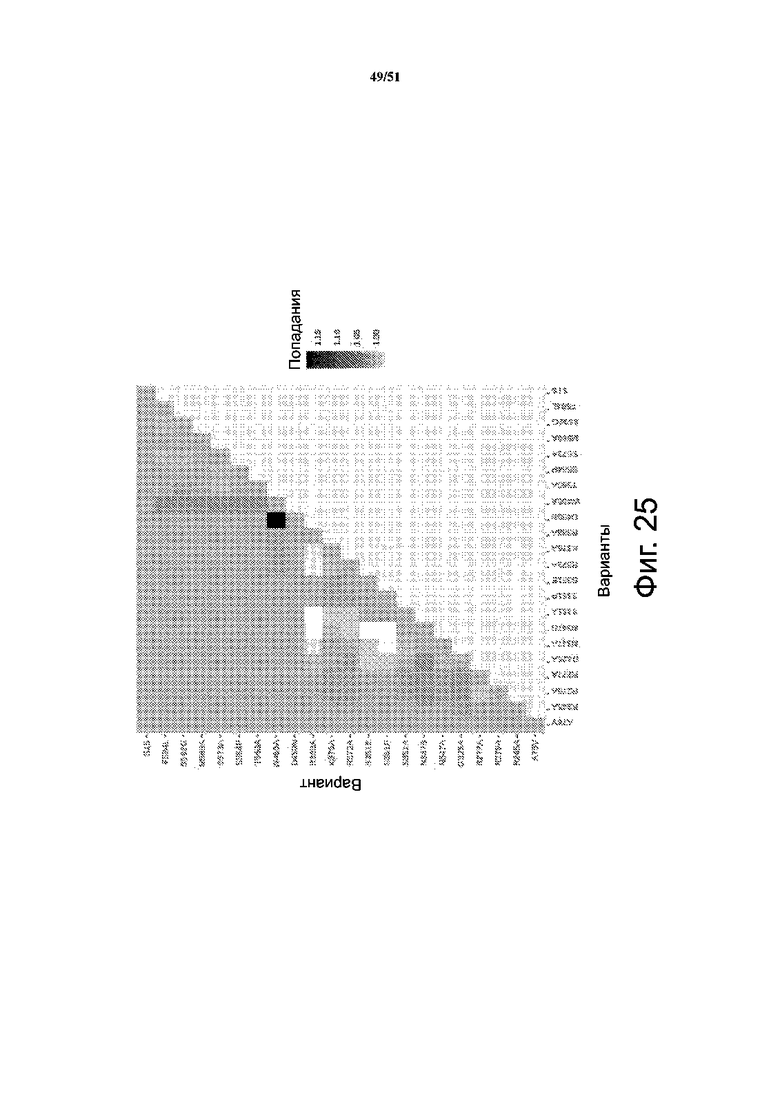
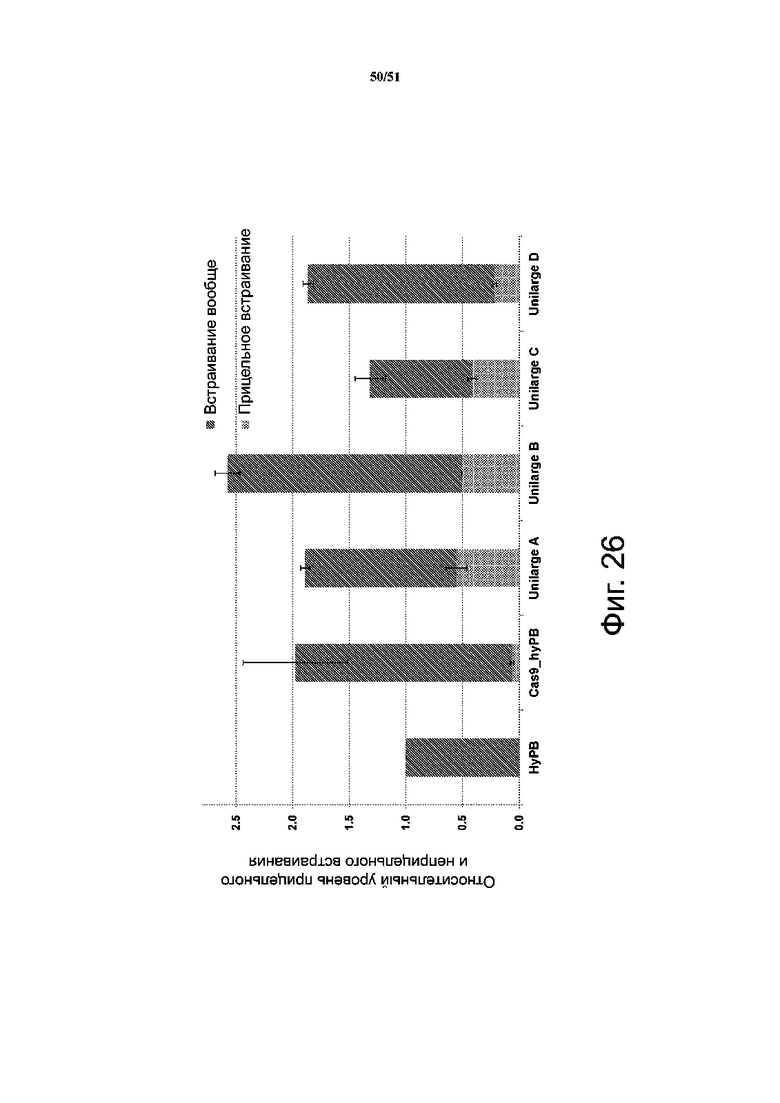

Изобретение относится к области биотехнологии, а именно к конструкции из нуклеиновой кислоты, включающей последовательность первого полинуклеотида, содержащего нуклеиновую кислоту, кодирующую первый ДНК-связывающий белок, сконструированный для связывания с определенной последовательностью геномной ДНК в геноме, причем первый ДНК-связывающий белок представляет собой белок Cas9 и последовательность второго полинуклеотида, содержащего нуклеиновую кислоту, кодирующую второй ДНК-связывающий белок, который обеспечивает встраивание экзогенной нуклеиновой кислоты в геном, причем второй ДНК-связывающий белок представляет собой модифицированную гиперактивную PiggyBac с улучшенной специфичностью встраивания экзогенной нуклеиновой кислоты в геном по сравнению с гиперактивной PiggyBac, а также к вектору, клетке и композиции, ее содержащим. Изобретение эффективно для контролируемого сайт-специфичного встраивания одной копии или нескольких копий последовательности экзогенной нуклеиновой кислоты в клетки. 5 н. и 18 з.п. ф-лы, 27 ил., 12 табл., 21 пр.
1. Конструкция из нуклеиновой кислоты, включающая:
a) последовательность первого полинуклеотида, содержащего нуклеиновую кислоту, кодирующую первый ДНК-связывающий белок, сконструированный для связывания с определенной последовательностью геномной ДНК в геноме, причем первый ДНК-связывающий белок представляет собой белок Cas9;
b) последовательность второго полинуклеотида, содержащего нуклеиновую кислоту, кодирующую второй ДНК-связывающий белок, который обеспечивает встраивание экзогенной нуклеиновой кислоты в геном, причем второй ДНК-связывающий белок представляет собой модифицированную гиперактивную PiggyBac с улучшенной специфичностью встраивания экзогенной нуклеиновой кислоты в геном по сравнению с гиперактивной PiggyBac; и
c) необязательно последовательность полинуклеотида, содержащего нуклеиновую кислоту, кодирующую линкер;
причем конструкция из нуклеиновой кислоты кодирует слитый белок, включающий первый ДНК-связывающий белок, второй ДНК-связывающий белок и необязательно линкер между первым ДНК-связывающим белком и вторым ДНК-связывающим белком; и
при этом слитый белок обеспечивает встраивание экзогенной нуклеиновой кислоты в определенный сайт генома,
при этом модифицированная гиперактивная транспозаза PiggyBac содержит мутации по одной или нескольким из числа аминокислот 245, 268, 275, 277, 287, 290, 315, 325, 341, 346, 347, 350, 351, 356, 357, 372, 375, 388, 409, 412, 432, 447, 450, 460, 461, 465, 517, 560, 564, 571, 573, 576, 586, 587, 589, 592 и 594 в соответствии с аминокислотной последовательностью гиперактивной PiggyBac по SEQ ID NO: 9.
2. Конструкция из нуклеиновой кислоты по п. 1, при этом белок Cas9 выбран из группы, состоящей из Cas9 человека, никазы Cas9 и «мертвого» Cas9.
3. Конструкция из нуклеиновой кислоты по любому из пп. 1, 2, при этом линкер включает последовательность XTEN или последовательность GGS.
4. Конструкция из нуклеиновой кислоты по любому из пп. 1-3, при этом 3'-конец последовательности первого полинуклеотида соединяется с 5'-концом второго полинуклеотида.
5. Конструкция из нуклеиновой кислоты по любому из пп. 1-4, при этом:
a) первый ДНК-связывающий белок представляет собой белок Cas9, а
b) второй ДНК-связывающий белок представляет собой модифицированную гиперактивную PiggyBac с улучшенной специфичностью встраивания экзогенной нуклеиновой кислоты в геном по сравнению с гиперактивной PiggyBac;
причем конструкция из нуклеиновой кислоты включает (c) последовательность полинуклеотида, содержащего нуклеиновую кислоту, кодирующую линкер, включающий последовательность XTEN или последовательность GGS, и
при этом 3'-конец последовательности первого полинуклеотида соединяется с 5'-концом второго полинуклеотида.
6. Конструкция из нуклеиновой кислоты по любому из пп. 1-5, при этом мутации модифицированной гиперактивной транспозазы PiggyBac включают одну или несколько модификаций аминокислот из числа R245A, D268N, R275A/R277A, K287A, K290A, K287A/K290A, R315A, G325A, R341A, D346N, N347A, N347S, T350A, S351E, S351P, S351A, K356E, N357A, R372A, K375A, R372A/K375A, R388A, K409A, K412A, K409A/K412A, K432A, D447A, D447N, D450N, R460A, K461A, R460A/K461A, W465A, S517A, T560A, S564P, S571N, S573A, K576A, H586A, I587A, M589V, S592G или F594L в соответствии с аминокислотной последовательностью гиперактивной PiggyBac по SEQ ID NO: 9.
7. Конструкция из нуклеиновой кислоты по любому из пп. 1-6, при этом модифицированная гиперактивная транспозаза PiggyBac содержит мутации по одной или нескольким из числа аминокислот 245, 275, 277, 325, 347, 351, 372, 375, 388, 450, 465, 560, 564, 573, 589, 592, 594 в соответствии с аминокислотной последовательностью гиперактивной PiggyBac по SEQ ID NO: 9.
8. Конструкция из нуклеиновой кислоты по п. 7, при этом мутации модифицированной гиперактивной транспозазы PiggyBac включают одну или несколько модификаций аминокислот из числа R245A, R275A, R277A, R275A/R277A, G325A, N347A, N347S, S351E, S351P, S351A, R372A, K375A, R388A, D450N, W465A, T560A, S564P, S573A, M589V, S592G или F594L в соответствии с аминокислотной последовательностью гиперактивной PiggyBac по SEQ ID NO: 9.
9. Конструкция из нуклеиновой кислоты по п. 7, при этом модифицированная гиперактивная транспозаза PiggyBac имеет аминокислотную последовательность по SEQ ID NO: 9, где:
i. в положении 245 находится аминокислота A,
ii. в положении 275 находится аминокислота R или A,
iii. в положении 277 находится аминокислота R или A,
iv. в положении 325 находится аминокислота A или G,
v. в положении 347 находится аминокислота N или A,
vi. в положении 351 находится аминокислота E, P или A,
vii. в положении 372 находится аминокислота R,
viii. в положении 375 находится аминокислота A,
ix. в положении 450 находится аминокислота D или N,
x. в положении 465 находится аминокислота W или A,
xi. в положении 560 находится аминокислота T или A,
xii. в положении 564 находится аминокислота P или S,
xiii. в положении 573 находится аминокислота S или A,
xiv. в положении 592 находится аминокислота G или S и
xv. в положении 594 находится аминокислота L или F.
10. Конструкция из нуклеиновой кислоты по п. 7, при этом модифицированная гиперактивная транспозаза PiggyBac имеет аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 120, 121, 122, 123, 124, 125, 126, 127, 128 и 129.
11. Конструкция из нуклеиновой кислоты по п. 7, при этом модифицированная гиперактивная транспозаза PiggyBac имеет аминокислотную последовательность, которая по меньшей мере на 80% идентична последовательности, выбранной из группы, состоящей из SEQ ID NO: 119, 120, 121, 122, 123, 124, 125, 126, 127, 128 и 129, причем модифицированная гиперактивная PiggyBac проявляет большую специфичность встраивания ДНК в геном по сравнению с гиперактивной PiggyBac.
12. Конструкция из нуклеиновой кислоты по любому из пп. 1-6, где указанная модифицированная гиперактивная транспозаза PiggyBac включает модификацию аминокислоты D450N в соответствии с нумерацией аминокислот по SEQ ID NO: 9; или где указанная модифицированная гиперактивная транспозаза PiggyBac включает модификации аминокислот R245A, G325A, и S573P, в соответствии с нумерацией аминокислот по SEQ ID NO: 9.
13. Конструкция из нуклеиновой кислоты по п. 12, где указанная модифицированная гиперактивная транспозаза PiggyBac включает модификацию аминокислоты D450N в соответствии с нумерацией аминокислот по SEQ ID NO: 9.
14. Конструкция из нуклеиновой кислоты по п. 13, где указанная модифицированная гиперактивная транспозаза PiggyBac включает модификации аминокислот R372A, K375A и D450N, в соответствии с нумерацией аминокислот по SEQ ID NO: 9.
15. Конструкция из нуклеиновой кислоты по п. 13, где указанная модифицированная гиперактивная транспозаза PiggyBac включает модификации аминокислот R245A, G325A, и S573P, в соответствии с нумерацией аминокислот по SEQ ID NO: 9.
16. Конструкция из нуклеиновой кислоты по п. 13, где указанная модифицированная гиперактивная транспозаза PiggyBac включает модификации аминокислот R245A и D450N, в соответствии с нумерацией аминокислот по SEQ ID NO: 9.
17. Конструкция из нуклеиновой кислоты по п. 15 или 16, где указанная модифицированная гиперактивная транспозаза PiggyBac включает модификации аминокислот R245A, G325A, D450N и S573P, в соответствии с нумерацией аминокислот по SEQ ID NO: 9.
18. Экспрессирующий вектор, содержащий конструкцию из нуклеиновой кислоты по любому из пп. 1-17, при этом экспрессирующий вектор подходит для экспрессии в клетках млекопитающих, дрожжевых клетках, клетках насекомых, клетках растений, грибковых клетках или клетках водорослей.
19. Клетка-хозяин для экспрессии конструкции из нуклеиновой кислоты по любому из пп. 1-17, содержащая конструкцию из нуклеиновой кислоты по любому из пп. 1-17 или экспрессирующий вектор по п. 18, где указанная конструкция из нуклеиновой кислоты, кодирующая слитый белок или экспрессирующий вектор предназначена для экспрессии конструкции из нуклеиновой кислоты в указанной клетке-хозяине.
20. Композиция, включающая конструкцию из нуклеиновой кислоты по любому из пп. 1-17, или экспрессирующий вектор по п. 18 и последовательность полинуклеотида, кодирующего экзогенную нуклеиновую кислоту для встраивания в геном, причем композиция содержится в упаковочном векторе или связана с ним, где указанная композиция предназначена для направленного редактирования нуклеиновых кислот.
21. Композиция по п. 20, при этом конструкция из нуклеиновой кислоты находится в виде РНК, ДНК или белка, а последовательность полинуклеотида, кодирующего экзогенную нуклеиновую кислоту, находится в виде ДНК или РНК.
22. Композиция по п. 20 или 21, при этом упаковочный вектор представляет собой наночастицы или лентивирусные частицы.
23. Способ контролируемого сайт-специфичного встраивания одной копии или нескольких копий последовательности экзогенной нуклеиновой кислоты в клетки, который включает:
a) введение в клетки конструкции из нуклеиновой кислоты по любому из пп. 1-17 или экспрессирующего вектора по п. 18; и
b) введение в клетки экзогенной нуклеиновой кислоты;
при этом связывание слитого белка с определенной последовательностью геномной ДНК в геноме клеток приводит к расщеплению генома и встраиванию одной или нескольких копий экзогенной нуклеиновой кислоты в геном этих клеток.
| WO 2018175872 A1, 27.09.2018 | |||
| WO 2016161207 A1, 06.10.2016 | |||
| WO 2004009792 A2, 29.01.2004 | |||
| CRAIG J | |||
| COATES et al | |||
| Site-directed genome modification: derivatives of DNA-modifying enzymes as targeting tools, TRENDS in Biotechnology, 2005, v | |||
| Прибор для равномерного смешения зерна и одновременного отбирания нескольких одинаковых по объему проб | 1921 |
|
SU23A1 |
| Топка с несколькими решетками для твердого топлива | 1918 |
|
SU8A1 |
| Ручная тележка для реклам | 1923 |
|
SU407A1 |
| А.А | |||
| ЧУГУНОВА и др | |||
| Методы изменения геномов: новая эра в молекулярной биологии, | |||
Авторы
Даты
2024-12-19—Публикация
2020-06-11—Подача