Перекрестная ссылка на связанные заявки
[0001] Настоящая заявка заявляет приоритет предварительной заявки США на патент 62/567750, поданной 3 октября 2017 г., «HPV-специфические связывающие молекулы», предварительной заявки США на патент 62/597411, поданной 11 декабря 2017 г., «HPV-специфические связывающие молекулы» и предварительной заявки США на патент 62/653529, поданной 5 апреля 2018 г., «HPV-специфические связывающие молекулы», содержание которых включено в качестве ссылки в полном объеме.
Включение путем ссылки на Список последовательностей
[0002] Настоящая заявка подается вместе со Списком последовательностей в электронном формате. Список последовательностей представлен в виде файла 735042014140SeqList.txt, созданного 28 сентября 2018 года, размер которого составляет 2288524 байта. Информация в электронном формате Списка последовательностей включена в качестве ссылки в полном объеме.
Область техники
[0003] Настоящее раскрытие относится в некоторых аспектах к связывающим молекулам, таким как те, которые распознают или связывают пептидный эпитоп вируса папилломы человека (HPV) 16 E6 или E7 в контексте молекулы главного комплекса гистосовместимости (MHC). В частности, настоящее раскрытие относится к T-клеточным рецепторам (TCR) или антителам, включая их антигенсвязывающие фрагменты, которые связывают или распознают пептидный эпитоп E6 HPV 16 или E7. Настоящее раскрытие дополнительно относится к сконструированным клеткам, содержащим такие связывающие молекулы, например TCR или антитела (и химерные антигенные рецепторы, содержащие антитела), и их применению в адоптивной клеточной терапии.
Уровень техники
[0004] Вирус папилломы человека (HPV) является распространенным вирусом среди людей, который в некоторых случаях может передаваться при контакте с кожей, и является распространенным вирусом, передаваемым половым путем. Определенные подтипы HPV, такие как HPV 16, могут привести к определенным видам злокачественных новообразований, таким как рак шейки матки и другие виды злокачественных новообразований. В некоторых случаях злокачественное новообразование может быть связано с экспрессией онкобелков HPV Е6 и/или Е7. Например, HPV Е6 и/или Е7 могут способствовать прогрессированию злокачественного новообразования путем нацеливания на сигнальные пути опухолевых супрессоров, которые участвуют в контроле клеточного роста. Доступны определенные терапевтические агенты, нацеленные на клетки или опухоли, экспрессирующие HPV 16, но необходимы улучшенные агенты против HPV 16. Представлены варианты осуществления, которые удовлетворяют таким потребностям.
Сущность изобретения
[0005] В настоящем описании представлены Т-клеточные рецепторы (TCR) или их антигенсвязывающие фрагменты. В некоторых вариантах осуществления TCR содержит альфа-цепь, содержащую вариабельную область альфа (Vα), и бета-цепь, содержащую вариабельную бета область (Vβ), где: область Vα содержит аминокислотную последовательность, представленную в любой из SEQ ID NO: 691, 709, 726, 741, 759, 775, 787, 799, 815, 830, 845, 857, 869, 881, 895, 908, 925, 937, 951, 963, 975, 987, 999, 999 или 1390, или аминокислотную последовательность, которая имеет с ними по меньшей мере 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% идентичности последовательности; и/или область Vβ содержит аминокислотную последовательность, представленную в любой из SEQ ID NO: 700, 718, 735, 750, 768, 781, 793, 808, 824, 839, 851, 863, 875, 887, 901, 917, 931, 945, 957, 969, 981, 993, 1008 или 1380, или аминокислотную последовательность, которая имеет с ними по меньшей мере 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% идентичности последовательности.
[0006] В некоторых вариантах осуществления область Vα содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность AX2X3X4X5X6X7X8X9X10X11X12X13X14 (SEQ ID NO: 1185), где X2 представляет собой A, G, V, Q, M или E; X3 представляет собой S, G, N, A, Y, R или P; X4 представляет собой E, S, A, G, F, N, D, V, P, L, I, M или R; X5 представляет собой R, N, H, T, D, G, S, P, L, Q или F; X6 представляет собой G, H, A, S, T или отсутствует; X7 представляет собой T, S, G или отсутствует; X8 представляет собой G или отсутствует; X9 представляет собой G, N, S или отсутствует; X10 представляет собой T, G, S, D, F, Y, A или N; X11 представляет собой Y, F, Q, R или N; X12 представляет собой K, Q или D; X13 представляет собой Y, L, T, M, F или V; X14 представляет собой I, T, S, R, Y или V; область Vα содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность X1X2X3X4X5X6X7X8X9X10KX12I (SEQ ID NO: 1186), где X1 представляет собой A или V; Х2 представляет собой А, V или Е; X3 представляет собой S, N, T, R или P; X4 представляет собой E, A, G, F, V, P, I, D или S; X5 представляет собой R, H, T, A P, S, G или F; X6 представляет собой G, H, L, T, S или A, отсутствует; X7 представляет собой S, T или отсутствует; X8 представляет собой G или отсутствует; X9 представляет собой G, T или отсутствует; Х10 представляет собой F, Y или N; X12 представляет собой Y, T или L; область Vα содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность AX2X3X4X5X6X7X8X9YKYI (SEQ ID NO: 1187), где X2 представляет собой A, V или E; X3 представляет собой S, N или R; X4 представляет собой E, G, V, P, I или D; X5 представляет собой R, T, P, S, G или F; X6 представляет собой G, T, S или отсутствует; X7 представляет собой S или отсутствует; X8 представляет собой G или отсутствует; Х9 представляет собой Т или отсутствует; область Vα содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность AX2X3X4X5X6X7X8X9X10X11X12X13X14 (SEQ ID NO: 1188), где X2 представляет собой G, V, Q или M; X3 представляет собой G, A, Y, S, N или R; X4 представляет собой S, G, L, I, M или R; Х5 представляет собой N, D, G, S, L, Q или R; X6 представляет собой A, S, G или отсутствует; X7 представляет собой G или отсутствует; X8 представляет собой G или отсутствует; X9 представляет собой G, N, S или отсутствует; X10 представляет собой S, D, Y, A, N или отсутствует; X11 представляет собой Y, Q или R; Х12 представляет собой К или Q; Х13 представляет собой L или V; X14 представляет собой S, T или V; область Vα содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность AX2X3X4X5X6X7X8X9X10X11X12X13T (SEQ ID NO: 1189), где X2 представляет собой G, V или Q; X3 представляет собой G, Y, S или N; Х4 представляет собой S, L или М; Х5 представляет собой N, G, L или R; X6 представляет собой A, S, G или отсутствует; X7 представляет собой G или отсутствует; X8 представляет собой G или отсутствует; X9 представляет собой G, S или отсутствует; X10 представляет собой S, Y, A, N или отсутствует; X11 представляет собой Y, Q или R; Х12 представляет собой К или Q; Х13 представляет собой L или V; область Vα содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность AX2X3X4X5X6X7YKLS (SEQ ID NO: 1190), где X2 представляет собой G или V; Х3 представляет собой А или Y; X4 представляет собой G, S или R; Х5 представляет собой D или S; X6 представляет собой N или отсутствует; X7 представляет собой D или отсутствует.
[0007] В некоторых вариантах осуществления область Vβ содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность AX2X3X4X5X6X7X8X9X10X11X12X13X14 (SEQ ID NO:1200), X2 представляет собой S, V, или I; X3 представляет собой S, N, или A; X4 представляет собой R, V, S, L, P, G, I, или A; X5 представляет собой F, G, Y, L, V, R, T, или S; X6 представляет собой L, G, A, D, R, V, или отсутствует; X7 представляет собой G, D, R, S, T, или отсутствует; X8 представляет собой S, или отсутствует; X9 представляет собой S, H, G, V, T, D, L, или отсутствует; X10 представляет собой T, S, A, G, P, N, или Y; X11 представляет собой D, Y, E, G, или N; X12 представляет собой T, E, G, или K; X13 представляет собой Q, Y, или L; X14 представляет собой Y, F, T, или I; область Vβ содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность ASSX4X5X6X7X8X9X10X11X12X13X14 (SEQ ID NO:1201), где X4 представляет собой R, V, S, L, G, или A; X5 представляет собой F, G, Y, L, V, T, или S; X6 представляет собой A, L, R, D, G, или отсутствует; X7 представляет собой G, D, T, или отсутствует; X8 представляет собой S, или отсутствует; X9 представляет собой S, H, G, T, D, L, или отсутствует; X10 представляет собой T, S, A, G, P, N, или Y; X11 представляет собой D, Y, E, G, или N; X12 представляет собой T, E, или G; X13 представляет собой Q, Y, или L; X14 представляет собой Y, F, или T; область Vβ содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность ASSX4X5X6X7X8X9X10TQY (SEQ ID NO: 1202), где X4 представляет собой R, L, или G; X5 представляет собой F, V, T, или Y; X6 представляет собой L, или A, отсутствует; X7 представляет собой G, или отсутствует; X8 представляет собой S, G, или отсутствует; X9 представляет собой T, G, P, или S; X10 представляет собой D, или E.
[0008] В настоящем описании представлены T-клеточные рецепторы (TCR) или их антигенсвязывающие фрагменты, содержащие альфа-цепь, содержащую вариабельную область альфа (Vα), и бета-цепь, содержащую вариабельную область бета (Vβ), где область Vα содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность AX2X3X4X5X6X7X8X9X10X11X12X13X14 (SEQ ID NO: 1185), где X2 представляет собой A, G, V, Q, M или E; X3 представляет собой S, G, N, A, Y, R или P; X4 представляет собой E, S, A, G, F, N, D, V, P, L, I, M или R; X5 представляет собой R, N, H, T, D, G, S, P, L, Q или F; X6 представляет собой G, H, A, S, T или отсутствует; X7 представляет собой T, S, G или отсутствует; X8 представляет собой G или отсутствует; X9 представляет собой G, N, S или отсутствует; X10 представляет собой T, G, S, D, F, Y, A или N; X11 представляет собой Y, F, Q, R или N; X12 представляет собой K, Q или D; X13 представляет собой Y, L, T, M, F или V; X14 представляет собой I, T, S, R, Y или V; область Vα содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность X1X2X3X4X5X6X7X8X9X10KX12I (SEQ ID NO: 1186), где X1 представляет собой A или V; Х2 представляет собой А, V или Е; X3 представляет собой S, N, T, R или P; X4 представляет собой E, A, G, F, V, P, I, D или S; X5 представляет собой R, H, T, A P, S, G или F; X6 представляет собой G, H, L, T, S или A, отсутствует; X7 представляет собой S, T или отсутствует; X8 представляет собой G или отсутствует; X9 представляет собой G, T или отсутствует; Х10 представляет собой F, Y или N; X12 представляет собой Y, T или L; область Vα содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность AX2X3X4X5X6X7X8X9YKYI (SEQ ID NO: 1187), где X2 представляет собой A, V или E; X3 представляет собой S, N или R; X4 представляет собой E, G, V, P, I или D; X5 представляет собой R, T, P, S, G или F; X6 представляет собой G, T, S или отсутствует; X7 представляет собой S или отсутствует; X8 представляет собой G или отсутствует; Х9 представляет собой Т или отсутствует; область Vα содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность AX2X3X4X5X6X7X8X9X10X11X12X13X14 (SEQ ID NO: 1188), где X2 представляет собой G, V, Q или M; X3 представляет собой G, A, Y, S, N или R; X4 представляет собой S, G, L, I, M или R; Х5 представляет собой N, D, G, S, L, Q или R; X6 представляет собой A, S, G или отсутствует; X7 представляет собой G или отсутствует; X8 представляет собой G или отсутствует; X9 представляет собой G, N, S или отсутствует; X10 представляет собой S, D, Y, A, N или отсутствует; X11 представляет собой Y, Q или R; Х12 представляет собой К или Q; Х13 представляет собой L или V; X14 представляет собой S, T или V; область Vα содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность AX2X3X4X5X6X7X8X9X10X11X12X13T (SEQ ID NO: 1189), где X2 представляет собой G, V или Q; X3 представляет собой G, Y, S или N; Х4 представляет собой S, L или М; Х5 представляет собой N, G, L или R; X6 представляет собой A, S, G или отсутствует; X7 представляет собой G или отсутствует; X8 представляет собой G или отсутствует; X9 представляет собой G, S или отсутствует; X10 представляет собой S, Y, A, N или отсутствует; X11 представляет собой Y, Q или R; Х12 представляет собой К или Q; Х13 представляет собой L или V; область Vα содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность AX2X3X4X5X6X7YKLS (SEQ ID NO: 1190), где X2 представляет собой G или V; Х3 представляет собой А или Y; X4 представляет собой G, S или R; Х5 представляет собой D или S; X6 представляет собой N или отсутствует; X7 представляет собой D или отсутствует.
[0009] В настоящем описании предложены T-клеточные рецепторы (TCR) или их антигенсвязывающие фрагменты, содержащие альфа-цепь, содержащую вариабельную область альфа (Vα), и бета-цепь, содержащую вариабельную область бета (Vβ), где: область Vβ содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность AX2X3X4X5X6X7X8X9X10X11X12X13X14 (SEQ ID NO:1200), X2 представляет собой S, V, или I; X3 представляет собой S, N, или A; X4 представляет собой R, V, S, L, P, G, I, или A; X5 представляет собой F, G, Y, L, V, R, T, или S; X6 представляет собой L, G, A, D, R, V, или отсутствует; X7 представляет собой G, D, R, S, T, или отсутствует; X8 представляет собой S, или отсутствует; X9 представляет собой S, H, G, V, T, D, L, или отсутствует; X10 представляет собой T, S, A, G, P, N, или Y; X11 представляет собой D, Y, E, G, или N; X12 представляет собой T, E, G, или K; X13 представляет собой Q, Y, или L; X14 представляет собой Y, F, T, или I; область Vβ содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность ASSX4X5X6X7X8X9X10X11X12X13X14 (SEQ ID NO:1201), где X4 представляет собой R, V, S, L, G, или A; X5 представляет собой F, G, Y, L, V, T, или S; X6 представляет собой A, L, R, D, G, или отсутствует; X7 представляет собой G, D, T, или отсутствует; X8 представляет собой S, или отсутствует; X9 представляет собой S, H, G, T, D, L, или отсутствует; X10 представляет собой T, S, A, G, P, N, или Y; X11 представляет собой D, Y, E, G, или N; X12 представляет собой T, E, или G; X13 представляет собой Q, Y, или L; X14 представляет собой Y, F, или T; область Vβ содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность ASSX4X5X6X7X8X9X10TQY (SEQ ID NO: 1202), где X4 представляет собой R, L, или G; X5 представляет собой F, V, T, или Y; X6 представляет собой L, или A, отсутствует; X7 представляет собой G, или отсутствует; X8 представляет собой S, G, или отсутствует; X9 представляет собой T, G, P, или S; X10 представляет собой D, или E.
[0010] В настоящем описании предложены Т-клеточные рецепторы (TCR) или их антигенсвязывающие фрагменты, содержащие альфа-цепь, содержащую вариабельную область альфа (Vα), и бета-цепь, содержащую вариабельную бета область (Vβ), где: область Vα содержит область 3, определяющую комплементарность, (CDR-3), представленную в любой из SEQ ID NO: 694, 712, 729, 744, 762, 776, 788, 802, 818, 832, 846, 858, 870, 882, 896, 911, 926, 940, 952, 964, 976, 988, 1002 или в последовательности, которая имеет с ними по меньшей мере 60%, 65%, 70%, 75%, 80%, 85%, 90% или 95% идентичности последовательности; область Vβ содержит область 3, определяющую комплементарность, (CDR-3), представленную в любой из SEQ ID NO: 703, 721, 736, 753, 769, 782, 794, 809, 825, 840, 852, 864, 876, 888, 902, 919, 932, 946, 958, 970, 982, 994 или 1010 или в последовательности, которая имеет с ними по меньшей мере 60%, 65%, 70%, 75%, 80%, 85%, 90% или 95% идентичности последовательности.
[0011] В некоторых вариантах осуществления область Vα содержит: область 1, определяющую комплементарность (CDR-1), содержащую аминокислотную последовательность X1X2X3X4X5X6 (SEQ ID NO: 1191), где X1 представляет собой N, S, D, T или V; X2 представляет собой S, V, R, T или I; Х3 представляет собой М, F, G, S, N, А, L, V или Р; X4 представляет собой F, S, N, A или отсутствует; X5 представляет собой D, S, Q, Y, N, V, T или P; и X6 представляет собой Y, S, R, N, G или T; и/или область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность X1X2X3X4X5X6X7X8 (SEQ ID NO: 1192), где X1 представляет собой I, V, L, G, N, T, Y или M; X2 представляет собой S, V, Y, L, P, F, I или T; X3 представляет собой S, Y, K, L, T или F; X4 представляет собой I, G, N, A, S или отсутствует; X5 представляет собой S, D или отсутствует; X6 представляет собой K, G, N, S, D, T или E; X7 представляет собой D, E, G, A, K, L или N; и X8 представляет собой K, V, D, P, N, T, L или M.
[0012] В некоторых вариантах осуществления область Vβ содержит область 1, определяющую комплементарность, (CDR-1), содержащую аминокислотную последовательность SX2X3X4X5 (SEQ ID NO: 1203), где X2 представляет собой G или N; Х3 представляет собой Н или D; Х4 представляет собой Т, L, N или V; и X5 представляет собой A, S, Y или T; и/или область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность X1X2X3X4X5X6 (SEQ ID NO: 1204), где X1 представляет собой F или Y; Х2 представляет собой Q, Y или N; X3 представляет собой G, N, R или Y; X4 представляет собой N, G, E или T; X5 представляет собой S, E, A или G; и X6 представляет собой A, E, I или Q.
[0013] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент связывает или распознает пептидный эпитоп вируса папилломы человека (HPV) 16 E7 в контексте молекулы MHC, пептидный эпитоп представляет собой или содержит E7 (11-19) YMLDLQPET (SEQ ID NO:236).
[0014] В некоторых вариантах осуществления TCR, предложенных в настоящем описании, область Vα содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 694, 712, 729, 744, 762, 776, 788, 802, 818, 832, 846, 858, 870, 882, 896, 911, 926, 940, 952, 964, 976, 988 или 1002, или CDR3, содержащуюся в аминокислотной последовательности, представленной в любой из SEQ ID NO: 691, 709, 726, 741, 759, 775, 787, 799, 815, 830, 845, 857, 869, 881, 895, 908, 925, 937, 951, 963, 975, 987 или 999; и/или область Vβ содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 703, 721, 736, 753, 769, 782, 794, 809, 825, 840, 852, 864, 876, 888, 902, 919, 932, 946, 958, 970, 982, 994, 1010 или 1381 или CDR3, содержащуюся в аминокислотной последовательности, представленной в любой из SEQ ID NO: 700, 718, 735, 750, 768, 781, 793, 808, 824, 839, 851, 863, 875, 887, 901, 917, 931, 931, 945, 957, 969, 981, 993, 1008 или 1380.
[0015] В некоторых вариантах осуществления область Vα дополнительно содержит: область 1, определяющую комплементарность (CDR-1), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 692, 710, 727, 742, 760, 171, 800, 816, 570, 909, 938, 151 или 1000; и/или область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 693, 711, 728, 743, 761, 172, 801, 817, 831, 571, 910, 939 152 или 1001.
[0016] В некоторых вариантах осуществления область Vβ содержит: область 1, определяющую комплементарность, (CDR-1), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 701, 719, 154, 751 или 139; и/или область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 702, 720, 155, 752, 140 или 918.
[0017] В некоторых вариантах осуществления, область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 692, 693 и 694, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 701, 702 и 703, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 710, 711 и 712, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 719, 720 и 721, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 727, 728 и 729, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 736, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 742, 743 и 744, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 751, 752 и 753, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 760, 761 и 762, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 719, 720 и 769, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 171, 172 и 776, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 782, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 742, 743 и 788, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 139, 140 и 794, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 800, 801 и 802, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 751, 752 и 809, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 816, 817 и 818, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 825, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 816, 831 и 832, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 840, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 171, 172 и 846, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 852, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 816, 831and 858, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 864, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 727, 728 и 870, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 876, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 570, 571 и 882, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 719, 720 и 888, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 816, 817 и 896, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 701, 702 и 902, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 909, 910 и 911, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 701, 702 и 919, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 727, 728 и 926, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 932, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 938, 939 и 940, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 946, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 727, 728 и 952, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 958, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 151,152 и 964, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 719, 720 и 970, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 727, 728 и 976, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 982, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 710, 711 и 988, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 719, 729 и 994, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 1000, 1001 и 1002, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 139, 1009 и 1010, соответственно.
[0018] В некоторых вариантах осуществления область Vα содержит область 1, определяющую комплементарность, (CDR-1), CDR-2 и CDR-3, соответственно, содержащие аминокислотные последовательности CDR-1, CDR-2 и CDR-3, содержащиеся в аминокислотной последовательности области Vα, представленные в любой из SEQ ID NO: 691, 709, 726, 741, 759, 775, 787, 799, 815, 830, 845, 857, 869, 881, 895, 908, 925, 937 951, 963, 975, 987, 999 или 1390; и/или область Vβ содержит область 1, определяющую комплементарность, (CDR-1), CDR-2 и CDR-3, соответственно, содержащие аминокислотные последовательности CDR-1, CDR-2 и CDR-3, содержащиеся в аминокислотной последовательности области Vβ, представленные в любой из SEQ ID NO: 700, 718, 735, 750, 768, 781, 793, 808, 824, 839, 851, 863, 875, 887, 901, 917, 931, 945, 957, 969, 981, 993, 1008 или 1380.
[0019] В некоторых вариантах осуществления, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 691 и 700, соответственно; области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 709 и 718, соответственно; области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO:726 и 735, соответственно; области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO:741 и 750, соответственно; области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO:759 и 768, соответственно; области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO:775 и 781, соответственно; области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO:787 и 793, соответственно; области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO:799 и 808, соответственно; области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO:815 и 824, соответственно; области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO:830 и 839, соответственно; области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO:845 и 851, соответственно; области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO:857 и 863, соответственно; области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO:869 и 875, соответственно; области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO:881 и 887, соответственно; области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO:895 и 901, соответственно; области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO:908 и 917, соответственно; области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO:925 и 931, соответственно; области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO:937 и 945, соответственно; области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO:951 и 957, соответственно; области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO:963 и 969, соответственно; области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO:975 и 981, соответственно; области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO:987 и 993, соответственно; области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO:999 и 1008, соответственно; или области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO:1390 и 1380, соответственно.
[0020] В некоторых вариантах осуществления альфа-цепь дополнительно содержит константную область альфа (Cα) и/или бета-цепь дополнительно содержит константную область бета (Cβ).
[0021] В некоторых вариантах осуществления области Cα и Cβ представляют собой константные области мыши. В некоторых вариантах осуществления область Cα содержит аминокислотную последовательность, представленную в SEQ ID NO: 262, 833, 1012, 1014, 1015, 1017, 1018, 1362, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности; и/или область Cβ содержит аминокислотную последовательность, представленную в SEQ ID NO: 263, 1013 или 1016, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности.
[0022] В некоторых вариантах осуществления области Cα и Cβ представляют собой константные области человека. В некоторых вариантах осуществления область Cα содержит аминокислотную последовательность, представленную в любой из SEQ ID NO: 212, 213, 215, 217, 218, 220 или 524, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности; и/или область Cβ содержит аминокислотную последовательность, представленную в любой из SEQ ID NO: 214, 216, 631 или 889, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности.
[0023] В некоторых вариантах осуществления а) альфа-цепь содержит: аминокислотную последовательность, представленную в любой из SEQ ID NO: 687, 705, 722, 737, 755, 771, 783, 795, 811, 826, 841, 853, 865, 877, 891, 904, 921, 933, 947 959, 971, 983, 995, 1386, последовательности аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности; или аминокислотную последовательность, кодируемую нуклеотидной последовательностью, представленной в любой из SEQ ID NO: 1049, 1051, 1055, 1057, 1059, 1061, 1063, 1065, 1067, 1069, 1071, 1073, 1075, 1075, 1077, 1079, 1081, 1083, 1085, 1087, 1089, 1091, или нуклеотидной последовательностью, которая имеет с ними по меньшей мере 90% идентичности последовательности; и/или бета-цепь содержит: аминокислотную последовательность, представленную в любой из SEQ ID NO: 696, 714, 731, 746, 764, 777, 789, 804, 820, 835, 847, 859, 871, 883, 897, 913, 927, 941, 953, 965, 977, 989, 1004 или 1376, последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности; или аминокислотную последовательность, кодируемую нуклеотидной последовательностью, представленной в SEQ ID NO: 1050, 1052, 1056, 1058, 1060, 1062, 1064, 1066, 1068, 1070, 1072, 1074, 1076, 1078, 1080, 1082, 1084, 1086, 1088, 1090 или 1092, или нуклеотидной последовательностью, которая имеет с ними по меньшей мере 90% идентичности последовательности.
[0024] В некоторых вариантах осуществления альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 687 и 696, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 705 и 714, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 722 и 731, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 737 и 746, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 755 и 764, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 771 и 777, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 783 и 789, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 795 и 804, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 811 и 820, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 826 и 835, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 841 и 847, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 853 и 859, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 865 и 871, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 877 и 883, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 891 и 897, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 904 и 913, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 921 и 927, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 933 и 941, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 947 и 953, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 959 и 965, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 971 и 977, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 983 и 989, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 995 и 1004, соответственно; или альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 1386 и 1376, соответственно.
[0025] В некоторых вариантах осуществления TCR или антигенсвязывающий фрагмент содержит одну или более модификаций в α-цепи и/или β-цепи, так что, когда TCR или его антигенсвязывающий фрагмент экспрессируются в клетке, частота ошибочного спаривания между α-цепью и β-цепью TCR и α-цепью и β-цепью эндогенного TCR уменьшается, экспрессия α-цепи и β-цепи TCR увеличивается и/или повышается стабильность α-цепи и β-цепи TCR, каждая по сравнению с экспрессией в клетке TCR или его антигенсвязывающего фрагмента, не содержащих одной или более модификаций. В некоторых вариантах осуществления одна или более модификаций представляют собой замену, делецию или вставку одной или более аминокислот в области Cα и/или области Cβ. В некоторых вариантах осуществления одна или более модификаций содержат замену(замены) для введения одного или более остатков цистеина, которые способны образовывать одну или более ненативных дисульфидных связей между альфа-цепью и бета-цепью.
[0026] В некоторых вариантах осуществления TCR содержит область Cα, содержащую цистеин в положении, соответствующем положению 48 с нумерацией, представленной в SEQ ID NO: 212, 213, 217, 218 или 524, или в положении, соответствующем положению 49 с нумерацией, представленной в SEQ ID NO: 215 или 220; и/или область Cβ, содержащую цистеин в положении, соответствующем положению 57 с нумерацией, представленной в SEQ ID NO: 214 или 216, или в положении, соответствующем положению 58 с нумерацией, представленной в SEQ ID NO: 631 или 889. В некоторых вариантах осуществления область Cα содержит аминокислотную последовательность, представленную в любой из SEQ ID NO: 196, 198, 200, 201, 203 или 525, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности, содержащие один или более остатков цистеина, способных к образованию ненативной дисульфидной связи с бета-цепью; и/или область Cβ содержит аминокислотную последовательность, представленную в любой из SEQ ID NO: 197,199, 632 или 890, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности, которые содержит один или более остатков цистеина, способных к образованию ненативной дисульфидной связи с альфа-цепью.
[0027] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент кодируются нуклеотидной последовательностью, которая была оптимизирована по кодонам. В некоторых вариантах осуществления а) альфа-цепь содержит: аминокислотную последовательность, представленную в любой из SEQ ID NO: 688, 706, 723, 738, 756, 772, 784, 796, 812, 827, 842, 854, 866, 878, 892, 905, 922, 934, 948, 960, 972, 984, 996 или 1387, последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности; или аминокислотную последовательность, кодируемую нуклеотидной последовательностью, представленной в любой из SEQ ID NO: 1129, 1131, 1133, 1135, 1137, 1139, 1141, 1143, 1145, 1147, 1149, 1151, 1153, 1155, 1157, 1159, 1161, 1163, 1165, 1167, 1169, 1171, 1173 или 1385, или нуклеотидной последовательностью, которая имеет с ними по меньшей мере 90% идентичности последовательности; и/или бета-цепь содержит: аминокислотную последовательность, представленную в любой из SEQ ID NO: 697, 715, 732, 747, 765, 778, 790, 805, 821, 836, 848, 860, 872, 884, 898, 914, 928, 942, 954, 966, 978, 990, 1005 или 1377, последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности; или аминокислотную последовательность, кодируемую нуклеотидной последовательностью, представленной в SEQ ID NO: 1130, 1132, 1134, 1136, 1138, 1140, 1142, 1144, 1146, 1148, 1150, 1152, 1154, 1156, 1158, 1160, 1162, 1164, 1166, 1168, 1170, 1172, 1174 или 1375, или нуклеотидной последовательности, которая имеет с ними по меньшей мере 90% идентичности последовательности.
[0028] В некоторых вариантах осуществления альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 688 и 697, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 706 и 715, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 723 и 732, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 738 и 747, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 756 и 765, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 772 и 778, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 784 и 790, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 796 и 805, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 812 и 821, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 827 и 836, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 842 и 848, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 854 и 860, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 866 и 872, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 878 и 884, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 892 и 898, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 905 и 914, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 922 и 928, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 934 и 942, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 948 и 954, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 960 и 966, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 972 и 978, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 984 и 990, соответственно; или альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 996 и 1005, соответственно; или альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 1387 и 1377, соответственно.
[0029] В некоторых вариантах осуществления альфа- и/или бета-цепь дополнительно содержит сигнальный пептид.
[0030] В некоторых вариантах осуществления альфа-цепь содержит сигнальный пептид, содержащий аминокислотную последовательность, представленную в любой из SEQ ID NO: 181, 184, 187, 189, 190, 192, 193, 310, 311; и/или бета-цепь содержит сигнальный пептид, содержащий аминокислотную последовательность, представленную в любой из SEQ ID NO: 182, 185, 186, 188, 191 или 194.
[0031] В некоторых вариантах осуществления представленный TCR или его антигенсвязывающий фрагмент выделены или очищены или являются рекомбинантными. В некоторых вариантах осуществления представленный TCR или его антигенсвязывающий фрагмент являются человеческими. В некоторых вариантах осуществления представленный TCR или его антигенсвязывающий фрагмент являются моноклональными. В некоторых вариантах осуществления представленный TCR или его антигенсвязывающий фрагмент являются одноцепочечными. В некоторых вариантах осуществления представленный TCR или его антигенсвязывающий фрагмент содержат две цепи.
[0032] В некоторых вариантах осуществления представленного TCR или его антигенсвязывающего фрагмента антигенспецифичность является, по меньшей мере, частично CD8-независимой.
[0033] В некоторых вариантах осуществления представленного TCR или его антигенсвязывающего фрагмента молекула MHC представляет собой молекулу HLA-A2.
[0034] Также в настоящем описании представлены молекулы нуклеиновой кислоты, кодирующие любой из TCR или его антигенсвязывающего фрагмента, описанных в настоящем документе, или их альфа- или бета-цепь.
[0035] В некоторых вариантах осуществления молекула нуклеиновой кислоты содержит нуклеотидную последовательность, кодирующую альфа-цепь, и/или нуклеотидную последовательность, кодирующую бета-цепь, где: нуклеотидная последовательность, кодирующая альфа-цепь, содержит последовательность, представленную в любой из SEQ ID NO: 1049, 1051, 1055, 1057, 1059, 1061, 1063, 1065, 1067, 1069, 1071, 1073, 1075, 1077, 1079, 1081, 1083, 1085, 1087, 1089, 1091, или в нуклеотидной последовательности, которая имеет с ними по меньшей мере 90% идентичность последовательности; нуклеотидная последовательность, кодирующая бета-цепь, содержит последовательность, представленную в SEQ ID NO: 1050, 1052, 1056, 1058, 1060, 1062, 1064, 1066, 1068, 1070, 1072, 1074, 1076, 1078, 1080, 1082, 1084, 1086, 1088, 1090 или 1092, или нуклеотидной последовательности, которая имеет с ними по меньшей мере 90% идентичности последовательности.
[0036] В некоторых вариантах осуществления нуклеотидная последовательность оптимизирована по кодонам.
[0037] В некоторых вариантах осуществления молекула нуклеиновой кислоты содержит нуклеотидную последовательность, кодирующую альфа-цепь, и/или нуклеотидную последовательность, кодирующую бета-цепь, где: нуклеотидная последовательность, кодирующая альфа-цепь, содержит последовательность, представленную в любой из SEQ ID NO: 1129, 1131, 1133, 1135, 1137, 1139, 1141, 1143, 1145, 1147, 1149, 1151, 1153, 1155, 1157, 1159, 1161, 1163, 1165, 1167, 1169, 1171, 1173 или 1385 или нуклеотидной последовательности, которая имеет с ними по меньшей мере 90% идентичности последовательности; нуклеотидная последовательность, кодирующая бета-цепь, содержит последовательность, представленную в SEQ ID NO: 1130, 1132, 1134, 1136, 1138, 1140, 1142, 1144, 1146, 1148, 1150, 1152, 1154, 1156, 1158, 1160, 1162, 1164, 1166, 1168, 1170, 1172, 1174 или 1375, или нуклеотидной последовательности, которая имеет с ними по меньшей мере 90% идентичности последовательности.
[0038] В некоторых вариантах осуществления нуклеотидная последовательность, кодирующая альфа-цепь, и нуклеотидная последовательность, кодирующая бета-цепь, разделены пептидной последовательностью, которая вызывает пропуск рибосомы. В некоторых вариантах осуществления пептид, который вызывает пропуск рибосомы, представляет собой пептид P2A или T2A и/или содержит последовательность аминокислот, представленную в SEQ ID NO: 204 или 211.
[0039] В некоторых вариантах осуществления молекула нуклеиновой кислоты содержит нуклеотидную последовательность, представленную в любой из SEQ ID NO: 448, 449, 450, 451, 452, 453, 454, 455, 456, 457, 458, 459, 460, 461, 462. 463, 464, 465, 466, 467, 468, 469, 470, 471, 472 или 1382, или нуклеотидной последовательности, имеющей с ними по меньшей мере 90% идентичности последовательности.
[0040] В настоящем описании также представлены Т-клеточные рецепторы (TCR) или их антигенсвязывающие фрагменты, содержащие альфа-цепь, содержащую вариабельную область альфа (Vα), и бета-цепь, содержащую вариабельную область бета (Vβ), где: область Vα содержит аминокислотную последовательность, представленную в любой из SEQ ID NO: 477, 492, 504, 510, 522, 536, 554, 569, 587, 599, 611, 623, 637, 649, 661 или 676, или аминокислотная последовательность, которая имеет с ними по меньшей мере 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% идентичности последовательности; и/или область Vβ содержит аминокислотную последовательность, представленную в любой из SEQ ID NO: 483, 498, 516, 530, 545, 560, 578, 593, 605, 617, 629, 643, 655, 667 или 685, или аминокислотную последовательность, которая имеет с ними по меньшей мере 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% идентичности последовательности.
[0041] В некоторых вариантах осуществления область Vα содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность AX2RX4AX6NNDMR, где X2 представляет собой V или M; Х4 представляет собой Р или D; и X6 представляет собой N или R (SEQ ID NO: 1221). В некоторых вариантах осуществления область Vβ содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность ASSX4WGX7SNQPX12H, где X4 представляет собой L, F или P; X7 представляет собой R или Q; и X12 представляет собой Q или L (SEQ ID NO: 1216); или область Vβ содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность ASSX4X5X6X7X8SGNTIY, где X4 представляет собой L или R; Х5 представляет собой W или Q; X6 представляет собой G или P; Х7 представляет собой R или S; и X8 представляет собой S или отсутствует (SEQ ID NO: 1217).
[0042] В настоящем описании также представлены Т-клеточные рецепторы (TCR) или их антигенсвязывающие фрагменты, содержащие альфа-цепь, содержащую вариабельную область альфа (Vα), и бета-цепь, содержащую вариабельную бета область (Vβ), где область Vα содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность AX2RX4AX6NNDMR, где X2 представляет собой V или M; Х4 представляет собой Р или D; и X6 представляет собой N или R (SEQ ID NO: 1221).
[0043] В настоящем описании также представлены Т-клеточные рецепторы (TCR) или их антигенсвязывающие фрагменты, содержащие альфа-цепь, содержащую вариабельную область альфа (Vα), и бета-цепь, содержащую вариабельную область бета (Vβ), где: область Vβ содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность ASSX4WGX7SNQPX12H, где X4 представляет собой L, F или P; X7 представляет собой R или Q; и X12 представляет собой Q или L (SEQ ID NO: 1216); или область Vβ содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность ASSX4X5X6X7X8SGNTIY, где X4 представляет собой L или R; Х5 представляет собой W или Q; X6 представляет собой G или P; Х7 представляет собой R или S; и X8 представляет собой S или отсутствует (SEQ ID NO: 1217).
[0044] В настоящем описании также представлены Т-клеточные рецепторы (TCR) или их антигенсвязывающие фрагменты, содержащие альфа-цепь, содержащую вариабельную область альфа (Vα), и бета-цепь, содержащую вариабельную бета область (Vβ), где: область Vα содержит область 3, определяющую комплементарность, (CDR-3), представленную в любой из SEQ ID NO: 478, 493, 505, 511, 523, 539, 555, 572, 588, 600, 612, 624, 638, 650, 662 или 679, или в последовательности, которая имеет с ними по меньшей мере 60%, 65%, 70%, 75%, 80%, 85%, 90% или 95% идентичности последовательности; область Vβ содержит область 3, определяющую комплементарность, (CDR-3), представленную в любой из SEQ ID NO: 486, 499, 517, 531, 548, 563, 581, 594, 606, 618, 630, 644, 656, 670 или 686, или в последовательности, которая имеет с ними по меньшей мере 60%, 65%, 70%, 75%, 80%, 85%, 90% или 95% идентичности последовательности.
[0045] В любом из предложенных в настоящем описании вариантов осуществления область Vα содержит: область 1, определяющую комплементарность (CDR-1), содержащую аминокислотную последовательность X1X2X3X4X5X6 (SEQ ID NO: 1191), где X1 представляет собой N, S, D, T, или V; X2 представляет собой S, V, R, T или I; Х3 представляет собой М, F, G, S, N, А, L, V или Р; X4 представляет собой F, S, N, A или отсутствует; X5 представляет собой D, S, Q, Y, N, V, T или P; и X6 представляет собой Y, S, R, N, G или T; и/или область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность X1X2X3X4X5X6X7X8 (SEQ ID NO: 1192), где X1 представляет собой I, V, L, G, N, T, Y или M; X2 представляет собой S, V, Y, L, P, F, I или T; X3 представляет собой S, Y, K, L, T или F; X4 представляет собой I, G, N, A, S или отсутствует; X5 представляет собой S, D или отсутствует; X6 представляет собой K, G, N, S, D, T или E; X7 представляет собой D, E, G, A, K, L или N; и X8 представляет собой K, V, D, P, N, T, L или M.
[0046] В любом из предложенных в настоящем описании вариантов осуществления область Vβ содержит: область 1, определяющую комплементарность, (CDR-1), содержащую аминокислотную последовательность SX2X3X4X5 (SEQ ID NO: 1203), где X2 представляет собой G или N; Х3 представляет собой Н или D; Х4 представляет собой Т, L, N или V; и X5 представляет собой A, S, Y или T; и/или область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность X1X2X3X4X5X6 (SEQ ID NO: 1204), где X1 представляет собой F или Y; Х2 представляет собой Q, Y или N; X3 представляет собой G, N, R или Y; X4 представляет собой N, G, E или T; X5 представляет собой S, E, A или G; и X6 представляет собой A, E, I или Q.
[0047] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент связывает или распознает пептидный эпитоп вируса папилломы человека (HPV) 16 E6 в контексте молекулы MHC, пептидный эпитоп представляет собой или содержит E6 (29-38) TIHDIILECV (SEQ ID NO: 233).
[0048] В некоторых вариантах осуществления область Vα содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 478, 493, 505, 511, 523, 539, 555, 572, 588, 600, 612, 624, 638, 650, 662 или 679, или CDR3, содержащуюся в аминокислотной последовательности, представленной в любой из SEQ ID NO: 477, 492, 504, 510, 522, 536, 554, 569, 587, 599, 611, 623, 637, 649, 661 или 676; и/или область Vβ содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 486, 499, 517, 531, 548, 563, 581, 594, 606, 618, 630, 644, 656, 670 или 686 или CDR3, содержащуюся в аминокислотной последовательности, представленной в любой из SEQ ID NO: 483, 498, 516, 530, 545, 560, 578, 593, 605, 617, 629, 643, 655, 667 или 685.
[0049] В некоторых вариантах осуществления область Vα также содержит: область 1, определяющую комплементарность (CDR-1), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 136, 161, 165, 537, 570, 142, 171 или 677; и/или область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 137, 162, 166, 538, 571, 143, 172 или 678.
[0050] В некоторых вариантах осуществления область Vβ содержит: область 1, определяющую комплементарность, (CDR-1), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 484, 148, 546, 561, 579, 168, 668 или 154; и/или область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 485, 149, 547, 562, 580, 169, 669 или 155.
[0051] В некоторых вариантах осуществления область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 136, 137 и 478, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 484, 485 и 486, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 161, 162 и 493, соответственно, а область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 499, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 165, 166 и 505, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 499, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 161, 162 и 511, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 517, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 136, 137 и 523, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 531, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 537, 538 и 539, соответственно, а область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащий аминокислотные последовательности SEQ ID NO: 546, 547 и 548, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 136, 137 и 555, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 561, 562 и 563, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 570, 571 и 572, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 579, 580 и 581, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 136, 137 и 600, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 594, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 136, 137 и 600, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 606, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 136, 137 и 612, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 618, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 136, 137 и 624, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 168, 169 и 630, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 142, 143 и 638, соответственно, а область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 561, 562 и 644, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 171, 172 и 650, соответственно, а область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 656, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 136, 137 и 662, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 668, 669 и 670, соответственно; или область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 677, 678 и 679, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 686, соответственно.
[0052] В некоторых вариантах осуществления область Vα содержит область 1, определяющую комплементарность, (CDR-1), CDR-2 и CDR-3, соответственно, содержащие аминокислотные последовательности CDR-1, CDR-2 и CDR-3, содержащиеся в аминокислотной последовательности области Vα, представленные в любой из SEQ ID NO: 477, 492, 504, 510, 522, 536, 554, 569, 587, 599, 611, 623, 637, 649, 661 или 676; и/или область Vβ содержит область 1, определяющую комплементарность, (CDR-1), CDR-2 и CDR-3, соответственно, содержащие аминокислотные последовательности CDR-1, CDR-2 и CDR-3, содержащиеся в аминокислотной последовательности области Vβ, представленные в любой из SEQ ID NO: 483, 498, 516, 530, 545, 560, 578, 593, 605, 617, 629, 643, 655, 667 или 685.
[0053] В некоторых вариантах осуществления область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 136, 137 и 478, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 484, 485 и 486, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 161, 162 и 493, соответственно, а область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 499, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 165, 166 и 505, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 499, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 161, 162 и 511, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 517, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 136, 137 и 523, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 531, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 537, 538 и 539, соответственно, а область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащий аминокислотные последовательности SEQ ID NO: 546, 547 и 548, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 136, 137 и 555, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 561, 562 и 563, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 570, 571 и 572, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 579, 580 и 581, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 136, 137 и 600, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 594, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 136, 137 и 600, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 606, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 136, 137 и 612, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 618, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 136, 137 и 624, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 168, 169 и 630, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 142, 143 и 638, соответственно, а область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 561, 562 и 644, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 171, 172 и 650, соответственно, а область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 656, соответственно; область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 136, 137 и 662, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 668, 669 и 670, соответственно; или область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 677, 678 и 679, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 686, соответственно.
[0054] В некоторых вариантах осуществления альфа-цепь дополнительно содержит константную область альфа (Cα) и/или бета-цепь дополнительно содержит константную область бета (Cβ).
[0055] В некоторых вариантах осуществления области Cα и Cβ представляют собой константные области мыши.
[0056] В некоторых вариантах осуществления область Cα содержит аминокислотную последовательность, представленную в SEQ ID NO: 262, 833, 1012, 1014, 1015, 1017, 1018 или 1362, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности; и/или область Cβ содержит аминокислотную последовательность, представленную в SEQ ID NO: 263, 1013 или 1016, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности.
[0057] В некоторых вариантах осуществления области Cα и Cβ представляют собой константные области человека. В некоторых вариантах осуществления область Cα содержит аминокислотную последовательность, представленную в любой из SEQ ID NO: 212, 213, 215, 217, 218, 220 или 524, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности; и/или область Cβ содержит аминокислотную последовательность, представленную в любой из SEQ ID NO: 214, 216, 631 или 889, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности.
[0058] В некоторых вариантах осуществления а) альфа-цепь содержит: аминокислотную последовательность, представленную в любой из SEQ ID NO: 473, 488, 500, 506, 518, 532, 550, 565, 583, 595, 607, 619, 633, 645, 657 или 672, последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности; или аминокислотную последовательность, кодируемую нуклеотидной последовательностью, представленной в любой из SEQ ID NO: 389, 430, 1019, 1021, 1023, 1025, 1027, 1029, 1031, 1033, 1035, 1037, 1039, 1041, 1043 или 1045, или нуклеотидной последовательностью, которая имеет с ними по меньшей мере 90% идентичности последовательности; и/или бета-цепь содержит: аминокислотную последовательность, представленную в любой из SEQ ID NO: 479, 494, 512, 526, 541, 556, 574, 589, 601, 613, 625, 639, 651, 663 или 681, последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности; или аминокислотную последовательность, кодируемую нуклеотидной последовательностью, представленной в SEQ ID NO: 390, 431, 1020, 1022, 1024, 1026, 1028, 1030, 1032, 1034, 1036, 1038, 1040, 1042, 1044 или 1046, или нуклеотидной последовательностью, которая имеет с ними по меньшей мере 90% идентичности последовательности.
[0059] В некоторых вариантах осуществления альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 473 и 479, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 488 и 494, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 500 и 494, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 506 и 512, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 518 и 526, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 532 и 541, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 550 и 556, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 565 и 574, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 583 и 589, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 595 и 601, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 607 и 613, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 619 и 625, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 633 и 639, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 645 и 651, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 657 и 663, соответственно; или альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 672 и 681, соответственно.
[0060] В некоторых вариантах осуществления TCR или антигенсвязывающий фрагмент содержит одну или более модификаций в α-цепи и/или β-цепи, так что, когда TCR или его антигенсвязывающий фрагмент экспрессируются в клетке, частота ошибочного спаривания между α-цепью и β-цепью TCR и α-цепью и β-цепью эндогенного TCR уменьшается, экспрессия α-цепи и β-цепи TCR увеличивается и/или повышается стабильность α-цепи и β-цепи TCR, каждая по сравнению с экспрессией в клетке TCR или его антигенсвязывающего фрагмента, не содержащих одной или более модификаций. В некоторых вариантах осуществления одна или более модификаций представляют собой замену, делецию или вставку одной или более аминокислот в области Cα и/или области Cβ. В некоторых вариантах осуществления одна или более модификаций содержат замену(замены) для введения одного или более остатков цистеина, которые способны образовывать одну или более ненативных дисульфидных связей между альфа-цепью и бета-цепью. В некоторых вариантах осуществления TCR содержит область Cα, содержащую цистеин, в положении, соответствующем положению 48 с нумерацией, представленной в SEQ ID NO: 212, 213, 217, 218 или 524, или в положении, соответствующем положению 49 с нумерацией, представленной в SEQ ID NO: 215 или 220; и/или область Cβ, содержащую цистеин, в положении, соответствующем положению 57 с нумерацией, представленной в SEQ ID NO: 214 или 216, или в положении, соответствующем положению 58 с нумерацией, представленной в SEQ ID NO: 631 или 889.
[0061] В некоторых вариантах осуществления область Cα содержит аминокислотную последовательность, представленную в любой из SEQ ID NO: 196, 198, 200, 201, 203 или 525, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности, содержащие один или более остатков цистеина, способных к образованию ненативной дисульфидной связи с бета-цепью; и/или область Cβ содержит аминокислотную последовательность, представленную в любой из SEQ ID NO: 197,199, 632 или 890, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности, которые содержит один или более остатков цистеина, способных к образованию ненативной дисульфидной связи с альфа-цепью.
[0062] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент кодируются нуклеотидной последовательностью, которая была оптимизирована по кодонам.
[0063] В некоторых вариантах осуществления а) альфа-цепь содержит: аминокислотную последовательность, представленную в любой из SEQ ID NO: 474, 489, 501, 507, 519, 533, 551, 566, 584, 596, 608, 620, 634, 646, 658 или 673, последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности; или аминокислотную последовательность, кодируемую нуклеотидной последовательностью, представленной в любой из SEQ ID NO: 1097, 1099, 1101, 1103, 1105, 1107, 1109, 1111, 1113, 1115, 1117, 1119, 1121, 1123, 1125 или 1127, или нуклеотидной последовательностью, которая имеет с ними по меньшей мере 90% идентичности последовательности; и/или бета-цепь содержит: аминокислотную последовательность, представленную в любой из SEQ ID NO: 480, 495, 513, 527, 542, 557, 575, 590, 602, 614, 626, 640, 652, 664 или 682, последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности; или аминокислотную последовательность, кодируемую нуклеотидной последовательностью, представленной в SEQ ID NO: 1098, 1100, 1102, 1104, 1106, 1108, 1110, 1112, 1114, 1116, 1118, 1120, 1122, 1124, 1126 или 1128, или нуклеотидной последовательностью, которая имеет с ними по меньшей мере 90% идентичности последовательности.
[0064] В некоторых вариантах осуществления альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 474 и 482, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 489 и 497, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 501 и 497, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 507 и 515, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 519 и 529, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 533 и 544, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 551 и 559, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 566 и 577, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 584 и 592, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 596 и 604, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 608 и 616, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 620 и 628, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 634 и 642, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 646 и 654, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 658 и 666, соответственно; или альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 673 и 684, соответственно.
[0065] В некоторых вариантах осуществления альфа- и/или бета-цепь дополнительно содержит сигнальный пептид. В некоторых вариантах осуществления альфа-цепь содержит сигнальный пептид, содержащий аминокислотную последовательность, представленную в любой из SEQ ID NO: 181, 184, 187, 189, 190, 192, 193, 310, 311; и/или бета-цепь содержит сигнальный пептид, содержащий аминокислотную последовательность, представленную в любой из SEQ ID NO: 182, 185, 186, 188, 191 или 194.
[0066] В некоторых вариантах осуществления представленный TCR или его антигенсвязывающий фрагмент выделены или очищены или являются рекомбинантными. В некоторых вариантах осуществления представленный TCR или его антигенсвязывающий фрагмент являются человеческими. В некоторых вариантах осуществления представленный TCR или его антигенсвязывающий фрагмент являются моноклональными. В некоторых вариантах осуществления представленный TCR или его антигенсвязывающий фрагмент являются одноцепочечными. В некоторых вариантах осуществления представленный TCR или его антигенсвязывающий фрагмент содержат две цепи.
[0067] В некоторых вариантах осуществления представленного TCR или его антигенсвязывающего фрагмента антигенспецифичность является, по меньшей мере, частично CD8-независимой.
[0068] В некоторых вариантах осуществления представленного TCR или его антигенсвязывающего фрагмента молекула MHC представляет собой молекулу HLA-A2.
[0069] Также в настоящем описании представлены молекулы нуклеиновой кислоты, кодирующие любой из TCR или его антигенсвязывающего фрагмента, описанных в настоящем документе, или их альфа- или бета-цепь.
[0070] В некоторых вариантах осуществления представленная молекула нуклеиновой кислоты содержит нуклеотидную последовательность, кодирующую альфа-цепь, и/или нуклеотидную последовательность, кодирующую бета-цепь, где: нуклеотидная последовательность, кодирующая альфа-цепь, содержит последовательность, представленную в любой из SEQ ID NO: 389., 430, 1019, 1021, 1023, 1025, 1027, 1029, 1031, 1033, 1035, 1037, 1039, 1041, 1043 или 1045, или нуклеотидной последовательности, которая имеет с ними по меньшей мере 90% идентичности последовательности; нуклеотидная последовательность, кодирующая бета-цепь, содержит последовательность, представленную в SEQ ID NO: 390, 431, 1020, 1022, 1024, 1026, 1028, 1030, 1032, 1034,1036, 1038, 1040, 1042, 1044 или 1046, или нуклеотидная последовательность, которая имеет с ними по меньшей мере 90% идентичности последовательности.
[0071] В некоторых вариантах осуществления нуклеотидная последовательность оптимизирована по кодонам.
[0072] В некоторых вариантах осуществления представленная молекула нуклеиновой кислоты содержит нуклеотидную последовательность, кодирующую альфа-цепь, и/или нуклеотидную последовательность, кодирующую бета-цепь, где: нуклеотидная последовательность, кодирующая альфа-цепь, содержит последовательность, представленную в любой из SEQ ID NOS: 1097, 1099, 1101, 1103, 1105, 1107, 1109, 1111, 1113, 1115, 1117, 1119, 1121, 1123, 1125 или 1127, или нуклеотидную последовательность, которая имеет с ними по меньшей мере 90% идентичности последовательности; нуклеотидная последовательность, кодирующая бета-цепь, содержит последовательность, представленную в SEQ ID NO: 1098, 1100, 1102, 1104, 1106, 1108, 1110, 1112, 1114, 1116, 1118, 1120, 1122, 1124, 1126 или 1128, или нуклеотидная последовательность, которая имеет с ними по меньшей мере 90% идентичности последовательности.
[0073] В некоторых вариантах осуществления нуклеотидная последовательность, кодирующая альфа-цепь, и нуклеотидная последовательность, кодирующая бета-цепь, разделены пептидной последовательностью, которая вызывает пропуск рибосомы. В некоторых вариантах осуществления пептид, который вызывает пропуск рибосомы, представляет собой пептид P2A или T2A и/или содержит последовательность аминокислот, представленную в SEQ ID NO: 204 или 211.
[0074] В некоторых вариантах осуществления представленная молекула нуклеиновой кислоты содержит нуклеотидную последовательность, представленную в любой из SEQ ID NO: 432, 433, 434, 435, 436, 437, 438, 439, 440, 441, 442, 443, 444, 445, 446 или 447, или нуклеотидной последовательности, имеющей с ними по меньшей мере 90% идентичности последовательности.
[0075] В некоторых вариантах нуклеиновая кислота является синтетической. В некоторых вариантах осуществления нуклеиновая кислота представляет собой кДНК.
[0076] В настоящем описании также представлены полинуклеотиды, содержащие (а) последовательность нуклеиновой кислоты, кодирующую любой из TCR или его антигенсвязывающую часть, представленные в настоящем описании, или содержащие молекулу нуклеиновой кислоты, кодирующую любой из представленного TCR или его антигенсвязывающего фрагмента, предложенных в настоящем описании и (b) одно или более плеч гомологии, связанных с последовательностью нуклеиновой кислоты, где одно или более плеч гомологии содержат последовательность, гомологичную одной или более областям открытой рамки считывания локуса константной области Т-клеточного рецептора альфа (TRAC).
[0077] Также в настоящем описании представлен полинуклеотид, содержащий (а) последовательность нуклеиновой кислоты, кодирующую часть Т-клеточного рецептора (TCR), причем указанная последовательность нуклеиновой кислоты, кодирующая (i) бета-цепь Т-клеточного рецептора (TCRβ), содержит вариабельную область бета (Vβ)) любого TCR или его антигенсвязывающего фрагмента, представленного в настоящем описании, и константную область бета (Cβ); и (ii) часть альфа-цепи Т-клеточного рецептора (TCRα), содержащую вариабельную область альфа (Vα) любого из TCR или его антигенсвязывающего фрагмента, представленного в настоящем описании, где часть цепи TCRα составляет менее чем полноразмерная цепь TCRα и (b) одно или более плеч гомологии, связанных с последовательностью нуклеиновой кислоты, где одно или более плеч гомологии содержат последовательность, гомологичную одной или более областям открытой рамки считывания локуса константной области Т-клеточного рецептора альфа (TRAC).
[0078] В некоторых вариантах осуществления любого из полинуклеотидов, предложенных в настоящем описании, цепь TCRα содержит константную область альфа (Cα), где по меньшей мере часть указанной Cα кодируется открытой рамкой считывания эндогенного локуса TRAC или его частичной последовательностью, когда TCR или его антигенсвязывающий фрагмент экспрессируется клеткой, в которую был введен полинуклеотид. В некоторых вариантах осуществления любого из полинуклеотидов, предложенных в настоящем описании, последовательность нуклеиновой кислоты (а) и один из одного или более плеч гомологии вместе содержат последовательность нуклеотидов, кодирующую Cα, которая менее чем полная длина нативной Cα, где по меньшей мере часть Cα кодируется открытой рамкой считывания эндогенного локуса TRAC или его частичной последовательностью, когда TCR или его антигенсвязывающий фрагмент экспрессируется клеткой, в которую был введен полинуклеотид. В некоторых вариантах осуществления последовательность нуклеиновой кислоты, кодирующая цепь TCRβ, находится в 5'-области от последовательности нуклеиновой кислоты, кодирующей часть цепи TCRα.
[0079] В некоторых вариантах осуществления любого из предложенных в настоящем описании полинуклеотидов последовательность нуклеиновой кислоты (а) не содержит интрона. В некоторых вариантах осуществления последовательность нуклеиновой кислоты (а) представляет собой последовательность, которая является экзогенной или гетерологичной для открытой рамки считывания эндогенного геномного локуса TRAC Т-клетки, необязательно Т-клетки человека. В некоторых вариантах осуществления последовательность нуклеиновой кислоты (а) находится в рамке с одним или более экзонами или их частичной последовательностью, необязательно экзоном 1 или его частичной последовательностью, открытой рамки считывания локуса TRAC, содержащейся в одном или более плечах гомологии. В некоторых вариантах осуществления часть Cα кодируется открытой рамкой считывания эндогенного локуса TRAC или его частичной последовательностью, а дополнительная часть Cα кодируется последовательностью нуклеиновой кислоты (а), где указанная дополнительная часть Cα меньше полной длины нативной Cα. В некоторых вариантах осуществления дополнительная часть Cα кодируется последовательностью нуклеотидов, начиная с остатка 3 и до остатка 3155 последовательности, представленной в SEQ ID NO: 348, или одним или более ее экзонами, или последовательностью, которая демонстрирует по меньшей мере 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичности последовательности с последовательностью нуклеотидов, начиная с остатка 3 и до остатка 3155 последовательности, представленной в SEQ ID NO: 348, или одним или более ее экзонами, или их частичной последовательностью. В некоторых вариантах осуществления дополнительная часть Cα кодируется последовательностью, представленной в SEQ ID NO: 1364, или последовательностью, которая демонстрирует, по меньшей мере, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичности последовательности с SEQ ID NO: 1364 или ее частичной последовательностью. В некоторых вариантах осуществления дополнительная часть области Cα и/или области Cβ, кодируемой последовательностью нуклеиновой кислоты (а), содержит одну или более модификаций, необязательно замену, делецию или вставку одной или более аминокислот по сравнению с нативной областью Cα и/или нативной областью Cβ, причем необязательно указанная одна или более модификаций вводят один или более остатков цистеина, которые способны образовывать одну или более ненативных дисульфидных связей между альфа-цепью и бета-цепью.
[0080] В некоторых вариантах осуществления любого из полинуклеотидов, предложенных в настоящем описании, одно или более плеч гомологии содержат 5'-плечо гомологии и/или 3'-плечо гомологии. В некоторых вариантах осуществления 5'-плечо гомологии содержит: а) последовательность, содержащую, по меньшей мере, содержащую или, по меньшей мере, 150, 200, 250, 300, 350, 400, 450, 500, 550 или 600 смежных нуклеотидов последовательности, которая демонстрирует по меньшей мере 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичности с последовательностью, представленной в SEQ ID NO: 1343; b) последовательность, содержащую, по меньшей мере, содержащую или, по меньшей мере 150, 200, 250, 300, 350, 400, 450, 500, 550 или 600 смежных нуклеотидов последовательности, представленной в SEQ ID NO: 1343; или c) последовательность, представленную в SEQ ID NO: 1343. В некоторых вариантах осуществления 3'-плечо гомологии содержит: а) последовательность, содержащую, по меньшей мере, содержащую или, по меньшей мере, 150, 200, 250, 300, 350, 400, 450, 500, 550 или 600 смежных нуклеотидов последовательности, которая демонстрирует по меньшей мере 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичности с последовательностью, представленной в SEQ ID NO: 1344; b) последовательность, содержащую, по меньшей мере, содержащую или, по меньшей мере, 150, 200, 250, 300, 350, 400, 450, 500, 550 или 600 смежных нуклеотидов последовательности, представленной в SEQ ID NO: 1344; или c) последовательность, представленную в SEQ ID NO: 1344.
[0081] В настоящем описании представлен полинуклеотид, содержащий (а) последовательность нуклеиновой кислоты, кодирующую любой из TCR или антигенсвязывающих фрагментов, предложенных в настоящем описании, или любую одну из молекул нуклеиновой кислоты, предложенных в настоящем описании, кодирующую TCR или его антигенсвязывающий фрагмент, и (b) одно или более плеч гомологии, связанных с последовательностью нуклеиновой кислоты, где одно или более плеч гомологии содержат последовательность, гомологичную одной или более областям открытой рамки считывания локуса константной области Т-клеточного рецептора бета (TRBC).
[0082] В настоящем описании представлен полинуклеотид, содержащий (а) последовательность нуклеиновой кислоты, кодирующую часть Т-клеточного рецептора (TCR), причем указанная последовательность нуклеиновой кислоты, кодирующая (i) альфа-цепь Т-клеточного рецептора (TCRα), содержит вариабельную область альфа (Vα) любого TCR или его антигенсвязывающего фрагмента, предложенных в настоящем описании, и константную область альфа (Cα); и (ii) часть бета-цепи Т-клеточного рецептора (TCRβ), содержащую вариабельную область бета (Vβ) любого из TCR или его антигенсвязывающего фрагмента, где часть цепи TCRβ меньше полноразмерной цепи TCRβ и (b) одно или более плеч гомологии, связанных с последовательностью нуклеиновой кислоты, где одно или более плеч гомологии содержат последовательность, гомологичную одной или более областям открытой рамки считывания локуса константной области Т-клеточного рецептора бета (TRBC).
[0083] В некоторых вариантах осуществления любого из представленных полинуклеотидов цепь TCRβ содержит константную область бета (Cβ), где, по меньшей мере, часть указанной Cβ кодируется открытой рамкой считывания эндогенного локуса TRBC или его частичной последовательностью, когда TCR или его антигенсвязывающий фрагмент экспрессируется клеткой, в которую был введен полинуклеотид. В некоторых вариантах осуществления последовательность нуклеиновой кислоты (a) и один из одного или более плеч гомологии вместе содержат последовательность нуклеотидов, кодирующих Cβ, которая меньше полной длины нативногой Cβ, где по меньшей мере часть Cβ кодируется открытой рамкой считывания эндогенного локуса TRAC или его частичной последовательности, когда TCR или его антигенсвязывающий фрагмент экспрессируется клеткой, в которую был введен полинуклеотид. В некоторых вариантах осуществления последовательность нуклеиновой кислоты, кодирующая цепь TCRα, находится в 5'-области от последовательности нуклеиновой кислоты, кодирующей часть цепи TCRα.
[0084] В некоторых вариантах осуществления любого из представленных полинуклеотидов последовательность нуклеиновой кислоты (а) не содержит интрон. В некоторых вариантах осуществления последовательность нуклеиновой кислоты (а) представляет собой последовательность, которая является экзогенной или гетерологичной для открытой рамки считывания эндогенного геномного локуса TRBC Т-клетки, необязательно Т-клетки человека. В некоторых вариантах осуществления последовательность нуклеиновой кислоты (а) находится в рамке с одним или более экзонами или их частичной последовательностью, необязательно экзоном 1 или его частичной последовательностью, открытой рамки считывания локуса TRBC, содержащейся в одном или более плечах гомологии. В некоторых вариантах осуществления часть Cβ кодируется открытой рамкой считывания эндогенного локуса TRBC или его частичной последовательностью, а дополнительная часть Cβ кодируется последовательностью нуклеиновой кислоты (а), где указанная дополнительная часть Cβ меньше полной длины нативной Cβ. В некоторых вариантах осуществления дополнительная часть Cβ кодируется последовательностью нуклеотидов, начиная с остатка 3 и до остатка 1445 последовательности, представленной в SEQ ID NO: 349, или одним или более ее экзонами, или последовательностью, которая демонстрирует по меньшей мере 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичности последовательности с последовательностью нуклеотидов, начиная с остатка 3 и до остатка 1445 последовательности, представленной в SEQ ID NO: 349, или одним или более ее экзонами, или их частичной последовательностью; или последовательностью нуклеотидов, начиная с остатка 3 и до остатка 1486 последовательности, представленной в SEQ ID NO: 1047, или одним или более ее экзонами, или последовательностью, которая демонстрирует, по меньшей мере, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичности последовательности с последовательностью нуклеотидов, начиная с остатка 3 и до остатка 1486 последовательности, представленной в SEQ ID NO: 1047, или одним или более ее экзонами, или их частичной последовательностью. В некоторых вариантах осуществления дополнительная часть области Cβ и/или Cα, кодируемой последовательностью нуклеиновой кислоты (а), содержит одну или более модификаций, необязательно замену, делецию или вставку одной или более аминокислот по сравнению с нативной областью Cβ и/или нативной областью Cα, причем необязательно указанная одна или более модификаций вводят один или более остатков цистеина, которые способны образовывать одну или более ненативных дисульфидных связей между альфа-цепью и бета-цепью.
[0085] В некоторых вариантах осуществления любого из представленных полинуклеотидов одно или более плеч гомологии содержат 5'-плечо гомологии и/или 3'-плечо гомологии.
[0086] В некоторых вариантах осуществления любого из представленных полинуклеотидов последовательность нуклеиновой кислоты (а) содержит один или более полицистронных элементов. В некоторых вариантах осуществления полицистронный элемент(элементы) расположен между последовательностью нуклеиновой кислоты, кодирующей TCRα или его часть, и последовательностью нуклеиновой кислоты, кодирующей TCRβ или его часть. В некоторых вариантах осуществления один или более полицистронных элементов находятся в 5'-области от последовательности нуклеиновой кислоты, кодирующей TCR или часть TCR, или молекулы нуклеиновой кислоты, кодирующей TCR. В некоторых вариантах осуществления один или более полицистронных элементов представляют собой или содержат последовательность пропуска рибосомы, необязательно, где последовательность пропуска рибосомы представляет собой элемент T2A, P2A, E2A или F2A.
[0087] В некоторых вариантах осуществления любого из представленных полинуклеотидов последовательность нуклеиновой кислоты (а) содержит один или более гетерологичных или регуляторных контрольных элементов, функционально связанных с контролем экспрессии TCR, когда он экспрессируется клеткой, в которую был введен полинуклеотид. В некоторых вариантах осуществления один или более гетерологичных регуляторных или контрольных элементов содержат промотор, энхансер, интрон, сигнал полиаденилирования, консенсусную последовательность Козака, акцепторную последовательность сплайсинга и/или донорную последовательность сплайсинга. В некоторых вариантах осуществления гетерологичный регуляторный или контрольный элемент содержит гетерологичный промотор, необязательно промотор фактора элонгации 1 альфа человека (EF1α) или промотор MND или его вариант.
[0088] В некоторых вариантах осуществления представленный полинуклеотид представляет собой линейный полинуклеотид, необязательно двухцепочечный полинуклеотид или одноцепочечный полинуклеотид.
[0089] В настоящем описании также представлены векторы, содержащие любую из молекул нуклеиновой кислоты, описанных в настоящем документе, или любой из полинуклеотидов, описанных в настоящем документе. В некоторых вариантах осуществления вектор представляет собой экспрессирующий вектор. В некоторых вариантах осуществления вектор представляет собой вирусный вектор. В некоторых вариантах осуществления вирусный вектор представляет собой ретровирусный вектор. В некоторых вариантах осуществления вирусный вектор представляет собой лентивирусный вектор. В некоторых вариантах осуществления лентивирусный вектор происходит из HIV-1. В некоторых вариантах осуществления любого из представленных векторов вирусный вектор представляет собой вектор AAV. В некоторых вариантах осуществления вектор AAV выбран из вектора AAV1, AAV2, AAV3, AAV4, AAV5, AAV6, AAV7 или AAV8.
[0090] В настоящем описании также представлены сконструированные клетки. В некоторых вариантах осуществления представленные сконструированные клетки содержат любую из молекул нуклеиновой кислоты, предложенных в настоящем описании, любой из полинуклеотидов, предложенных в настоящем описании, или любой из векторов, предложенных в настоящем описании.
[0091] В настоящем описании также представлены сконструированные клетки. В некоторых вариантах осуществления представленные сконструированные клетки содержат любой из TCR или его антигенсвязывающего фрагмента, описанных в настоящем документе.
[0092] В некоторых вариантах осуществления представленные сконструированные клетки содержат генетическое нарушение гена константной области Т-клеточного рецептора альфа (TRAC) и/или гена константной области Т-клеточного рецептора бета (TRBC). В некоторых вариантах осуществления ген TRBC представляет собой один или оба из гена константной области 1 Т-клеточного рецептора бета (TRBC1) или гена константной области 2 Т-клеточного рецептора бета (TRBC2).
[0093] В настоящем описании также представлены сконструированные клетки, содержащие TCR или его антигенсвязывающий фрагмент, необязательно рекомбинантный TCR или его антигенсвязывающий фрагмент, где: (1) клетка содержит генетическое нарушение гена константной области Т-клеточного рецептора альфа (TRAC) и/или гена константной области Т-клеточного рецептора бета (TRBC) и/или не экспрессирует, или не экспрессирует на детектируемом уровне, или экспрессирует менее чем 20%, 15%, 10%, 5%, 4%, 3%, 2% или 1% от уровня дикого типа, генный продукт эндогенного TRAC или TRBC; и (2) TCR или его антигенсвязывающий фрагмент или рекомбинантный TCR или его антигенсвязывающий фрагмент содержат любой TCR или его антигенсвязывающий фрагмент, представленный в настоящем описании, необязательно рекомбинантный TCR или антигенсвязывающий фрагмент. В некоторых вариантах осуществления сконструированная клетка содержит генетическое нарушение локуса константной области Т-клеточного рецептора (TRAC).
[0094] В некоторых вариантах осуществления любой из представленных сконструированных клеток эндогенный локус TRAC дополнительно модифицируется путем интеграции последовательности нуклеиновой кислоты, кодирующей любой из TCR или его антигенсвязывающего фрагмента, в локусе TRAC, необязательно посредством направляемой гомологией репарации (HDR). В некоторых вариантах осуществления эндогенный локус TRAC дополнительно модифицируется путем интеграции трансгенной последовательности, кодирующей часть TCR или его антигенсвязывающего фрагмента, необязательно посредством направляемой гомологией репарации (HDR).
[0095] В настоящем описании также представлена сконструированная клетка, содержащая модифицированный локус TRAC, кодирующий любой из TCR или его антигенсвязывающего фрагмента, предложенных в настоящем описании.
[0096] В настоящем описании также представлена сконструированная клетка, содержащая модифицированный локус TRAC, где эндогенный локус TRAC модифицируется путем интеграции трансгенной последовательности, кодирующей часть TCR, причем указанная трансгенная последовательность кодирует (i) бета-цепь Т-клеточного рецептора (TCRβ), содержащую вариабельную область бета (Vβ) любого из TCR или его антигенсвязывающего фрагмента и константную область бета (Cβ); и (ii) часть альфа-цепи Т-клеточного рецептора (TCRα), содержащую вариабельную область альфа (Vα) любого из TCR или его антигенсвязывающего фрагмента, где по меньшей мере часть константной области альфа (Cα) TCR кодируется открытой рамкой считывания эндогенного локуса TRAC или его частичной последовательностью.
[0097] В некоторых из любых вариантов осуществления представленных сконструированных клеток TCR или его антигенсвязывающий фрагмент содержит Cα, причем по меньшей мере часть указанной Cα кодируется открытой рамкой считывания или ее частичной последовательностью эндогенного локуса TRAC. В некоторых вариантах осуществления модифицированный локус TRAC содержит слияние в рамке (i) трансгенной последовательности, кодирующей часть TCR, и (ii) открытой рамки считывания или ее частичной последовательности эндогенного локуса TRAC. В некоторых вариантах осуществления трансгенная последовательность не содержит последовательности, кодирующей 3'-UTR или интрон. В некоторых вариантах осуществления открытая рамка считывания или ее частичная последовательность содержит 3'-UTR эндогенного локуса TRAC.
[0098] В некоторых из любых вариантов осуществления представленных сконструированных клеток трансгенная последовательность интегрирована в 3'-области от большинства 5'-нуклеотидов экзона 1 и в 5'-области от большинства 3'-нуклеотидов экзона 1 открытой рамки считывания эндогенного локуса TRAC. В некоторых вариантах осуществления по меньшей мере часть Cα кодируется по меньшей мере экзонами 2-4 открытой рамки считывания эндогенного локуса TRAC. В некоторых вариантах осуществления по меньшей мере часть Cα кодируется по меньшей мере частью экзона 1 и экзонов 2-4 открытой рамки считывания эндогенного локуса TRAC.
[0099] В некоторых вариантах осуществления любой из представленных сконструированных клеток трансгенная последовательность кодирует бета-цепь Т-клеточного рецептора (TCRβ) и/или вариабельную область альфа TCR (Vα).
[0100] В некоторых вариантах осуществления любой из представленных сконструированных клеток сконструированная клетка дополнительно содержит генетическое нарушение локуса константной области Т-клеточного рецептора бета (TRBC), необязательно локуса TRBC1 или TRBC2.
[0101] В некоторых вариантах осуществления любой из представленных сконструированных клеток сконструированная клетка содержит генетическое нарушение локуса константной области Т-клеточного рецептора бета (TRBC). В некоторых вариантах осуществления эндогенный локус TRBC дополнительно модифицируется путем интеграции последовательности нуклеиновой кислоты, кодирующей TCR или его антигенсвязывающий фрагмент, в локус TRBC, необязательно через HDR. В некоторых вариантах осуществления эндогенный локус TRBC дополнительно модифицируется путем интеграции трансгенной последовательности, кодирующей часть TCR или его антигенсвязывающий фрагмент, необязательно посредством репарации, направляемой гомологией (HDR).
[0102] В настоящем описании представлена сконструированная клетка, содержащая модифицированный локус TRBC, кодирующий любой из TCR или его антигенсвязывающего фрагмента.
[0103] В настоящем описании представлена сконструированная клетка, содержащая модифицированный локус TRBC, где эндогенный локус TRBC модифицируется путем интеграции трансгенной последовательности, кодирующей часть TCR, причем указанная трансгенная последовательность кодирует (i) альфа-цепь Т-клеточного рецептора (TCRα), содержащую вариабельную область альфа (Vα) любого из TCR или его антигенсвязывающего фрагмента и константную область альфа (Cα); и (ii) часть бета-цепи Т-клеточного рецептора (TCRβ), содержащую вариабельную область бета (Vβ) любого из TCR или его антигенсвязывающего фрагмента, где по меньшей мере часть константной области бета (Cβ) TCR кодируется открытой рамкой считывания эндогенного локуса TRBC или его частичной последовательностью.
[0104] В некоторых вариантах осуществления любой из представленных сконструированных клеток TCR или его антигенсвязывающий фрагмент содержит Cβ, причем по меньшей мере, часть указанной Cβ кодируется открытой рамкой считывания или ее частичной последовательностью эндогенного локуса TRBC. В некоторых вариантах осуществления модифицированный локус TRBC содержит слияние в рамке (i) трансгенной последовательности, кодирующей часть TCR, и (ii) открытой рамки считывания или ее частичной последовательности эндогенного локуса TRBC. В некоторых вариантах осуществления трансгенная последовательность не содержит последовательности, кодирующей 3'-UTR или интрон. В некоторых вариантах осуществления открытая рамка считывания или ее частичная последовательность содержит 3'-UTR эндогенного локуса TRBC. В некоторых вариантах осуществления трансгенная последовательность интегрирована в 3'-области от большинства 5'-нуклеотидов экзона 1 и в 5'-области от большинства 3'-нуклеотидов экзона 1 открытой рамки считывания эндогенного локуса TRBC. В некоторых вариантах осуществления по меньшей мере часть Cβ кодируется по меньшей мере экзонами 2-4 открытой рамки считывания эндогенного локуса TRBC. В некоторых вариантах осуществления по меньшей мере часть Cβ кодируется по меньшей мере частью экзона 1 и экзонов 2-4 открытой рамки считывания эндогенного локуса TRBC.
[0105] В некоторых вариантах осуществления любой из представленных сконструированных клеток трансгенная последовательность кодирует альфа-цепь Т-клеточного рецептора (TCRα) и/или вариабельную область бета TCR (Vβ).
[0106] В некоторых вариантах осуществления любой из представленных сконструированных клеток локус TRBC представляет собой один или оба из локуса константной области 1 Т-клеточного рецептора бета (TRBC1) или константной области 2 Т-клеточного рецептора бета (TRBC2). В некоторых вариантах осуществления сконструированная клетка дополнительно содержит генетическое нарушение локуса константной области Т-клеточного рецептора альфа (TRAC).
[0107] В некоторых вариантах осуществления любой из представленных сконструированных клеток трансгенная последовательность или последовательность нуклеиновой кислоты, кодирующая TCR или его антигенсвязывающий фрагмент, содержит один или более полицистронных элементов. В некоторых вариантах осуществления один или более полицистронных элементов находятся в 5'-области от трансгенной последовательности или последовательности нуклеиновой кислоты, кодирующей TCR или его антигенсвязывающий фрагмент. В некоторых вариантах осуществления полицистронный элемент(элементы) расположен между последовательностью нуклеиновой кислоты, кодирующей TCRα или его часть, и последовательностью нуклеиновой кислоты, кодирующей TCRβ или его часть. В некоторых вариантах осуществления один или более полицистронных элементов представляют собой или содержат последовательность пропуска рибосомы, необязательно, где последовательность пропуска рибосомы представляет собой элемент T2A, P2A, E2A или F2A.
[0108] В некоторых вариантах осуществления любой из представленных сконструированных клеток трансгенная последовательность или последовательность нуклеиновой кислоты, кодирующая TCR или его антигенсвязывающий фрагмент, содержит один или более гетерологичных или регуляторных контрольных элементов, функционально связанных с контролем экспрессии TCR, при экспрессии из сконструированной. В некоторых вариантах осуществления один или более гетерологичных регуляторных или контрольных элементов содержат промотор, энхансер, интрон, сигнал полиаденилирования, консенсусную последовательность Козака, акцепторную последовательность сплайсинга и/или донорную последовательность сплайсинга. В некоторых вариантах осуществления гетерологичный регуляторный или контрольный элемент содержит гетерологичный промотор, необязательно промотор фактора элонгации 1 альфа человека (EF1α) или промотор MND или его вариант.
[0109] В некоторых вариантах осуществления любой из представленных сконструированных клеток TCR или его антигенсвязывающий фрагмент является гетерологичным по отношению к клетке. В некоторых вариантах осуществления сконструированная клетка представляет собой клеточную линию. В некоторых вариантах осуществления сконструированная клетка представляет собой первичную клетку, полученную от пациента. В некоторых вариантах осуществления пациентом является млекопитающее. В некоторых вариантах осуществления пациентом является человек. В некоторых вариантах осуществления сконструированная клетка представляет собой Т-клетку. В некоторых вариантах осуществления Т-клетка представляет собой CD8+. В некоторых вариантах осуществления Т-клетка представляет собой CD4+.
[0110] В настоящем описании также представлены способы получения любых сконструированных клеток, описанных в настоящем документе, которые включают введение любого из векторов, описанных в настоящем документе, в клетку in vitro или ex vivo. В некоторых вариантах осуществления вектор представляет собой вирусный вектор, и введение осуществляется путем трансдукции.
[0111] В настоящем описании также представлен способ получения клетки, включающий введение молекулы нуклеиновой кислоты, кодирующей любой из TCR или его антигенсвязывающего фрагмента, предложенных в настоящем описании, любой из молекулы нуклеиновой кислоты, представленной в настоящем документе, любого из полинуклеотидов, предложенных в настоящем описании, или любого из векторов, предложенных в настоящем описании, в клетку in vitro или ex vivo.
[0112] В некоторых вариантах осуществления способы, представленные в настоящем описании, включают введение в клетку одного или более агентов, где каждый из одного или более агентов независимо способен индуцировать генетическое нарушение гена константной области Т-клеточного рецептора альфа (TRAC) и/или гена константной области Т-клеточного рецептора бета (TRBC). В некоторых вариантах осуществления один или более агентов, способных индуцировать генетическое нарушение, включают ДНК-связывающий белок или ДНК-связывающую нуклеиновую кислоту, которая специфически связывается или гибридизуется с сайтом-мишенью. В некоторых вариантах осуществления один или более агентов, способных вызывать генетическое нарушение, включают (а) слитый белок, содержащий ДНК-нацеленный белок и нуклеазу, или (b) РНК-направляемую нуклеазу. В некоторых вариантах осуществления ДНК-нацеленный белок или РНК-направленная нуклеаза включают белок цинкового пальца (ZFP), белок TAL или нуклеазу (Cas), ассоциированную с нуклеиновой кислотой CRISPR (короткие палиндромные повторы, регулярно расположенные группами), специфичную для сайта-мишени в пределах гена TRAC и/или TRBC. В некоторых вариантах осуществления один или более агентов включают нуклеазу цинкового пальца (ZFN), нуклеазу TAL-эффектора (TALEN) или комбинацию CRISPR-Cas9, которая специфически связывается, распознает или гибридизуется с сайтом-мишенью. В некоторых вариантах осуществления каждый из одного или более агентов содержит гид-РНК (гРНК), имеющую нацеливающий домен, который комплементарен по меньшей мере одному сайту-мишени.
[0113] В некоторых вариантах осуществления один или более агентов вводят в виде рибонуклеопротеинового комплекса (RNP), содержащего гРНК и белок Cas9. В некоторых вариантах осуществления RNP вводят посредством электропорации, генной пушки, трансфекции фосфатом кальция, сжатия или сдавливания клеток. В некоторых вариантах осуществления RNP вводят посредством электропорации.
[0114] В некоторых вариантах осуществления один или более агентов вводят в виде одного или более полинуклеотидов, кодирующих гРНК и/или белок Cas9.
[0115] В некоторых вариантах осуществления любого из представленных способов один или более агентов и молекулы нуклеиновой кислоты, полинуклеотида или вектора вводят одновременно или последовательно, в любом порядке. В некоторых вариантах осуществления молекулу нуклеиновой кислоты, полинуклеотид или вектор вводят после введения одного или более агентов. В некоторых вариантах осуществления молекулу нуклеиновой кислоты, полинуклеотид или вектор вводят сразу после или в течение примерно 30 секунд, 1 минуты, 2 минут, 3 минут, 4 минут, 5 минут, 6 минут, 6 минут, 8 минут, 9 минуты, 10 минут, 15 минут, 20 минут, 30 минут, 40 минут, 50 минут, 60 минут, 90 минут, 2 часов, 3 часов или 4 часов после введения агента.
[0116] В настоящем описании также представлены композиции. В некоторых вариантах осуществления композиции содержат любые из сконструированных клеток, описанных в настоящем документе. В некоторых вариантах осуществления сконструированные клетки содержат CD4+ и/или CD8+ T-клетки. В некоторых вариантах осуществления сконструированные клетки содержат CD4+ и CD8+ T-клетки.
[0117] В настоящем описании также представлены композиции. В некоторых вариантах осуществления композиции содержат любые сконструированные клетки CD8+ и любые сконструированные клетки CD4+, описанные в настоящем документе.
[0118] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент связывают или распознает пептидный эпитоп HPV 16 в контексте молекулы МНС, которая является по меньшей мере частично независимой от CD8. В некоторых вариантах осуществления CD8+ клетка и CD4+ клетка сконструированы с одним и тем же TCR или его антигенсвязывающим фрагментом и/или каждая из них сконструирована с TCR или его антигенсвязывающим фрагментом, которые связывают или распознают один и тот же пептидный эпитоп HPV 16 в контекст молекулы МНС.
[0119] В некоторых вариантах осуществления любая из предложенных в настоящем описании композиций также содержит фармацевтически приемлемое вспомогательное вещество.
[0120] В настоящем описании также представлены способы лечения. В некоторых вариантах осуществления представленные способы лечения включают введение любой из сконструированных клеток, описанных в настоящем документе, пациенту, имеющему заболевание или расстройство, ассоциированное с HPV.
[0121] В настоящем описании также представлены способы лечения. В некоторых вариантах осуществления представленные способы лечения включают введение любой композиции, описанной в настоящем документе, пациенту, имеющему заболевание или расстройство, ассоциированное с HPV. В некоторых вариантах осуществления заболевание или расстройство ассоциировано с HPV16. В некоторых вариантах осуществления заболевание или расстройство представляет собой злокачественное новообразование. В некоторых вариантах осуществления пациентом является человек.
[0122] В настоящем описании также представлены композиции, такие как любая из композиций, описанных в настоящем документе, для применения при лечении заболевания или расстройства, ассоциированного с HPV.
[0123] В настоящем описании также представлены применения композиций, таких как любая из предложенных в настоящем описании композиций, для изготовления лекарственного средства для лечения заболевания или расстройства, ассоциированного с HPV. В некоторых вариантах осуществления заболевание или расстройство ассоциировано с HPV16. В некоторых вариантах осуществления заболевание или расстройство представляет собой злокачественное новообразование. В некоторых вариантах осуществления пациентом является человек.
Краткое описание чертежей
[0124] ФИГ. 1 демонстрирует литическую активность моноклональных Т-клеточных линий, экспрессирующих иллюстративные TCR, инкубированные с клетками-мишенями SiHa или клетками-мишенями Caski, на основе процента клеток-мишеней, положительных по каспазе, в различные оценочные моменты времени. Конкретно, результаты показаны для Т-клеточных линий, экспрессирующих модифицированную версию TCR 5 и модифицированную версию TCR 12.
[0125] На ФИГ. 2A-2L показаны результаты проточной цитометрии для связывания тетрамера CD4+ клеточной линией, полученной из Jurkat (Отр. кнтрл. CD4+), и CD4+ клеточной линией, полученной из Jurkat, экспрессирующей различные E6(29-38)-специфичные TCR (CD4+ TCR-E6 (29)), CD4+ клеточной линией, полученной из Jurkat, которая также экспрессирует экзогенный CD8 (CD8), или CD4+ клеточной линией, полученной из Jurkat, которая также экспрессирует экзогенный CD8 и различные E6 (29-38) -специфичные TCR (CD8+ TCR-E6 (29)). В частности, результаты показаны для эталонного TCR, модифицированной версии TCR 5, модифицированной версии TCR 4, модифицированной версии TCR 3 и модифицированной версии TCR 8.
[0126] На ФИГ. 3А-3D показаны результаты проточной цитометрии для связывания тетрамера CD4+ клеточной линией, полученной из Jurkat (Отр. контрл. CD4+), клеточной линией, экспрессирующей различные E7 (11-19) -специфичные TCR (CD4+ TCR-E7 (11-19)), CD4+ клеточной линией, полученной из Jurkat, которая также экспрессирует экзогенный CD8 (CD8), или CD4+ клеточной линией, полученной из Jurkat, которая также экспрессирует экзогенный CD8 и различные E7 (11-19) -специфичные TCR (CD8+ TCR-E7) (11-19)). В частности, результаты показаны для модифицированной версии TCR 7 и модифицированной версии TCR 12.
[0127] На ФИГ. 4А-4В показаны результаты проточной цитометрии для связывания тетрамера CD4+ клеточной линией, полученной из Jurkat (Отр. кнтрл., CD4+), CD4+ клеточной линией, полученной из Jurkat, экспрессирующей различные специфичные для E7 (86-93) TCR (CD4+ TCR-E7 (86-93)), CD4+ клеточной линией, полученной из Jurkat, которая также экспрессирует экзогенный CD8 (CD8), или CD4+ клеточной линией, полученной из Jurkat, которая также экспрессирует экзогенный CD8 и различные E7 (86-93) -специфичные TCR (CD8+ TCR-E7(86-93)). В частности, результаты показаны для модифицированной версии TCR 11.
[0128] На ФИГ. 5А-5С показаны результаты проточной цитометрии для связывания тетрамера и в клеточной линии, полученной из Jurkat, которая также экспрессирует экзогенный CD8 и различные E6 (29-38) -специфичные TCR в CD8+ клетках. Результаты показаны для TCR 9, TCR13, TCR14, эталонного TCR, способного связываться с HLA-A2/E6 (29-38) (эталонный TCR) и клеток, которые были подвергнуты ложной трансфекции (имитация) (ФИГ.5A); TCR 17, TCR 21, TCR 22, эталонный TCR и Имитация (ФИГ. 5B); и TCR 18, TCR 23, TCR 24 и TCR 27 (ФИГ. 5C).
[0129] На ФИГ. 5D-5F показаны результаты проточной цитометрии для связывания тетрамера и в клеточной линии, полученной из Jurkat, которая также экспрессирует экзогенный CD8 и различные E6(29-38) -специфичные TCR. Результаты показаны для TCR 15, TCR 16, TCR 17, TCR 19, TCR 20 и TCR 21 (ФИГ. 5D); TCR 18, TCR 23, TCR 24, TCR 27 и TCR 28 (ФИГ. 5E); и TCR 25, TCR 26, TCR 29 и TCR 30 (ФИГ. 5F).
[0130] На ФИГ. 6А-6G показаны результаты проточной цитометрии для связывания тетрамера и в клеточной линии, полученной из Jurkat, которая также экспрессирует экзогенный CD8 и различные E7(11-19) -специфичные TCR. Результаты показаны для TCR 12 и клеток, которые были ложно трансфецированы (имитация) (ФИГ.6A); TCR 31, TCR 32, TCR 33 и TCR 34 (ФИГ. 6B); TCR 12, TCR 49, TCR 50 и TCR 51 (ФИГ.6C); TCR 35, TCR 36, TCR 37, TCR 38, TCR 53 и TCR 54 (ФИГ.6D); TCR 39, TCR 40, TCR 41, TCR 42, TCR 43 и TCR 44 (ФИГ. 6E); и TCR 45, TCR 46, TCR 47, TCR 48, TCR 54 и TCR 55 (ФИГ. 6F). На ФИГ.6G показаны соответствующие результаты проточной цитометрии для связывания тетрамера в клетках, которые сконструированы для экспрессии рекомбинантных TCR, которые, как было обнаружено, демонстрируют CD8-зависимое связывание тетрамера (слева, TCR49) или CD8-независимое связывание тетрамера (справа, TCR37).
[0131] На ФИГ.7А и 7В показана эффективность нокаута эндогенного гена TCR в первичных Т-клетках, измеренная путем оценки экспрессии CD3 с использованием проточной цитометрии.
[0132] На ФИГ.8А показаны результаты проточной цитометрии для оценки эффективности нокаута эндогенного TCR в клетках, электропорированных RNP, нацеленных на эндогенные гены TCR (эндо. TCR KO) по сравнению с контрольными клетками (эндо. TCR WT). На ФИГ.8B показаны результаты анализа проточной цитометрией, оценивающего экспрессию суррогатного маркера для экспрессии TCR и связывания тетрамера E6 в CD4 и CD8 клетках. На ФИГ.8С показана продукция IFNγ клетками с эндогенным TCR (эндо. TCR WT) и эндогенным TCR KO (эндо. TCR KO) для TCR 16 и 31, и сравнивали с контролем ложной трансдукции.
[0133] На ФИГ. 9A-9J показана экспрессия TCR, которую оценивали по связыванию тетрамера E7 (11-19), цитолитической активности и продукции интерферона-гамма после инкубации с антиген-специфическими клетками-мишенями, в клетках, сконструированных для экспрессии различных иллюстративных рекомбинантных TCR. На ФИГ.9А (TCR 49), ФИГ.9D (TCR 53) и ФИГ.9G (TCR 37) показана экспрессия TCR, которую оценивали по связыванию тетрамера E7 (11-19). На ФИГ.9B (TCR 49), ФИГ. 9E (TCR 53), ФИГ. 9H (TCR 37) и ФИГ. 9J (TCR 37) показана цитолитическая активность, которую отслеживали по уменьшенному световому сигналу NucRed. На ФИГ. 9C (TCR 49), ФИГ. 9F (TCR 53), ФИГ. 9I (TCR 37) и ФИГ. 9J (TCR 37) показана продукция интерферона-гамма TCR-экспрессирующими клетками после инкубации с антиген-специфическими клетками-мишенями.
[0134] На ФИГ.10А-10В показана экспрессия иллюстративных TCR в клетках с нокаутом эндогенных генов TCR по сравнению с клетками, которые сохранили эндогенные гены TCR, что оценивали по связыванию тетрамера E7 (11-19).
[0135] На ФИГ.11А-11В показана оценка чувствительности к пептиду иллюстративных TCR в клетках с нокаутом эндогенных генов TCR по сравнению с клетками, которые сохранили эндогенные гены TCR, что оценивали по продукции гамма-интерферона после инкубации с T2-клетками, стимулированными пептидом.
[0136] На ФИГ. 12А показано, что лизис клеток-мишеней измеряли по потере мишени каждые 2 часа с использованием визуализации живых клеток. На ФИГ. 12B-12C показана оценка цитолитической активности и продукции цитокина интерферона-гамма примерными TCR в клетках с нокаутом эндогенных генов TCR по сравнению с клетками, которые сохранили эндогенные гены TCR.
[0137] На ФИГ. 13 показаны изменения объема опухоли с течением времени на мышиной модели с подкожными опухолями UPCI: SCC152 (ATCC® CRL-3240 ™), где мышам вводили только CD4+ рекомбинантные TCR-экспрессирующие клетки (перевернутый треугольник), только CD8+ рекомбинантные TCR-экспрессирующие клетки (треугольник) или смесь CD4+ и CD8+ рекомбинантных TCR-экспрессирующих клеток (квадрат) по сравнению с мышами, которые не получали никакого лечения (круг).
[0138] На ФИГ. 14A-14G представляют собой изображения нескольких иллюстративных гРНК.
[0139] На ФИГ. 14А изображена модульная молекула гРНК, частично полученная (или частично смоделированная на основе последовательности) из Streptococcus pyogenes (S. pyogenes) в виде дуплексной структуры (SEQ ID NO: 42 и 43 Международной публикации РСТ. No. WO2015161276, соответственно в порядке появления);
[0140] На ФИГ. 14B изображена мономолекулярную (или химерная) молекула гРНК, частично полученная из S. pyogenes, в виде дуплексной структуры (SEQ ID NO: 44 Международной публикации РСТ. No. WO2015161276);
[0141] На ФИГ. 14C изображена мономолекулярная молекула гРНК, полученная частично из S. pyogenes, в виде дуплексной структуры (SEQ ID NO: 45 Международной публикации РСТ. No. WO2015161276);
[0142] На ФИГ. 14D изображена мономолекулярная молекула гРНК, частично полученная из S. pyogenes, в виде дуплексной структуры (SEQ ID NO: 46 Международной публикации РСТ No. WO2015161276);
[0143] На ФИГ. 14E изображена мономолекулярная молекула гРНК, частично полученная из S. pyogenes, в виде дуплексной структуры (SEQ ID NO: 47 Международной публикации РСТ. No. WO2015161276);
[0144] На ФИГ. 14F изображена модульная молекула гРНК, частично полученная из Streptococcus thermophilus (S. thermophilus) в виде дуплексной структуры (SEQ ID NO: 48 и 49 Международной публикации РСТ No. WO2015161276, соответственно в порядке появления);
[0145] На ФИГ. 14G показано выравнивание модульных молекул гРНК S. pyogenes и S. thermophiles (SEQ ID NO: 50-53 Международной публикации РСТ No. WO2015161276, соответственно в порядке появления).
[0146] На ФИГ. 15A-15G изображено выравнивание последовательностей Cas9 от Chylinski et al. (RNA Biol. 2013; 10 (5): 726-737). N-концевой RuvC-подобный домен помечен рамкой и обозначен «у». Два других RuvC-подобных домена помечены рамкой и обозначены «b». HNH-подобный домен помечен рамкой и обозначен «g». Sm: S. mutans (SEQ ID NO: 1331); Sp: S. pyogenes (SEQ ID NO: 1332); St: S. thermophilus (SEQ ID NO: 1333); Li: L. innocua (SEQ ID NO: 1334). Мотив: это мотив на основе четырех последовательностей: остатки, сохраненные во всех четырех последовательностях, указаны однобуквенным сокращением аминокислот; «*» обозначает любую аминокислоту, найденную в соответствующем положении любой из четырех последовательностей; и «-» обозначает любую аминокислоту, например любую из 20 встречающихся в природе аминокислот.
[0147] На ФИГ. 16A-16C изображено выравнивание последовательностей Cas9 из S. pyogenes и Neisseria meningitides (N. meningitidis). N-концевой RuvC-подобный домен помечен рамкой и обозначен «Y». Два других RuvC-подобных домена помечены рамкой и обозначены «B». HNH-подобный домен помечен рамкой и обозначен «G». Sp: S. pyogenes; Нм: N. meningitidis. Мотив: это мотив на основе двух последовательностей: остатки, консервативные в обеих последовательностях, обозначены одним аминокислотным обозначением; «*» обозначает любую аминокислоту, найденную в соответствующем положении любой из двух последовательностей; «-» обозначает любую аминокислоту, например любую из 20 встречающихся в природе аминокислот, а «-» обозначает любую аминокислоту, например любую из 20 встречающихся в природе аминокислот, или то, что она отсутствует.
[0148] На ФИГ. 17А изображена поверхностная экспрессия CD8 и тетрамера пептид-МНС в комплексе с антигеном, распознаваемым примерным рекомбинантным TCR (TCR 49), что оценивали с помощью проточной цитометрии, для Т-клеток, подверженных нокауту эндогенных генов, кодирующих TCR, сконструированных для экспрессии TCR 49 с использованием различных способов экспрессии: клетки, подвергали лентивирусной трансдукции для случайной интеграции рекомбинантных TCR-кодирующих последовательностей («TCR 49 Lenti»), клетки, подвергали случайной интеграции, и CRISPR/Cas9-опосредованному нокаут (KO) TRAC («TCR 49 Lenti KO»); или клетки, подвергали направленной интеграции с помощью HDR в локусе TRAC рекомбинантных TCR-кодирующих последовательностей, под контролем человеческого промотора EF1α (TCR 49 HDR KO). На ФИГ. 17B и 17C показана средняя интенсивность флуоресценции (MFI; ФИГ. 17B) и коэффициент вариации (стандартное отклонение сигнала в популяции клеток, деленное на среднее значение сигнала в соответствующей популяции; ФИГ. 17C) экспрессии на клеточной поверхности связывания тетрамера пептид-MHC в CD8+ T-клетках, сконструированных для экспрессии TCR 49.
[0149] На ФИГ. 18А-18С изображено окрашивание и плотность рецептора для TCR (TCR 49) с использованием антитела Vbeta22, специфичного для рекомбинантного TCR или тетрамера пептид-MHC.
[0150] На ФИГ. 19 показана средняя цитолитическая активность различных рекомбинантных TCR 49-экспрессирующих CD8+ T-клеток, как описано выше, полученных от 2 доноров, представленных площадью под кривой (AUC)% умерщвления, по сравнению с контролем ложной трансдукции и нормализованной к экспрессии Vbeta22 (специфическое окрашивание для рекомбинантного TCR) для каждой группы, описанной выше, после инкубации эффекторных клеток, как описано выше, с клетками-мишенями, экспрессирующими E7 HPV 16 при соотношении эффектор/мишень (E:T) 10:1, 5:1 и 2,5:1. CD8+ Клетки, трансдуцированные лентивирусом, кодирующим эталонный TCR, способный связываться с E7 HPV 16, но содержащий мышиные области Cα и Cβ, оценивали в качестве контроля («Lenti Ref»).
[0151] На ФИГ. 20 изображена средняя секреция IFNγ (пг/мл) различными рекомбинантными TCR 49-экспрессирующими CD8+ T-клетками, как описано выше.
[0152] На ФИГ. На ФИГ.21А и 21В показана поверхностная экспрессия CD8, CD3, Vbeta22 (специфическое окрашивание для рекомбинантного TCR) и тетрамера пептид-MHC в комплексе с антигеном, распознаваемым рекомбинантным TCR, согласно оценке проточной цитометрией для Т-клеток, подвергнутых нокауту эндогенных TCR-кодирующих генов, сконструированных для экспрессии рекомбинантного T-клеточного рецептора (TCR) с использованием различных методов экспрессии: клеток, подвергнутых CRISPR/Cas9-опосредованному нокауту (KO) TRAC и TRBC («TCRαβ KO») или сохраняющих экспрессию эндогенного TCR («TCRαβ WT»); клеток, подвергнутых направленной интеграции с помощью HDR в локус TRAC рекомбинантных TCR-кодирующих последовательностей, связанных с промотором EF1α или MND («HDR EF1α» или «HDR MND»); клеток, подвергнутых лентивирусной трансдукции для случайной интеграции рекомбинантных TCR-кодирующих последовательностей («lenti человек») или рекомбинантных TCR-кодирующих последовательностей, содержащих константный домен мыши («lenti мышь»), или ложной трансдукции в качестве контроля («ложн. трансд.»).
[0153] На ФИГ. 21C и 21D показана среднегеометрическая интенсивность флуоресценции (gMFI) экспрессии на клеточной поверхности Vbeta22 и связывания тетрамера пептид-MHC в CD8+ (ФИГ. 21C) или CD4+ (ФИГ. 21D) Т-клетках, сконструированных для экспрессии рекомбинантного Т-клеточного рецептора (TCR) с использованием различных способов экспрессии, как описано выше.
[0154] На ФИГ. 21E и 21F показан коэффициент вариации (стандартное отклонение сигнала в популяции клеток, деленное на среднее значение сигнала в соответствующей популяции) в CD8+ T-клетках, сконструированных для экспрессии рекомбинантного T-клеточного рецептора (TCR) с использованием различных методов экспрессии, как описано выше, для экспрессии тетрамера пептид-MHC (ФИГ. 21E) и связывания Vbeta22 (ФИГ. 21F).
[0155] На ФИГ. 22A-22C изображена поверхностная экспрессия CD3 и CD8, которую оценивали с помощью проточной цитометрии, для Т-клеток, подвергнутых нокауту эндогенных TCR-кодирующих генов, сконструированных для экспрессии рекомбинантного Т-клеточного рецептора (TCR) с использованием различных методов экспрессии: клетки, подвергнутые CRISPR/Cas9-опосредованному нокауту (KO) TRAC, TRBC или обоих TRAC и TRBC; клетки, подвергнутые направленной интеграции с помощью HDR в локусе TRAC рекомбинантных TCR-кодирующих последовательностей, связанных с промотором EF1α, промотором MND или эндогенным промотором TCR-альфа, с использованием последовательности пропуска рибосомы P2A («HDR EF1α», «HDR MND» или «HDR P2A», соответственно), или клетки, подвергнутые ложной трансдукции в качестве контроля («ложн. трансд.») (ФИГ. 22A); клетки, сохраняющие экспрессию эндогенного TCR и подлежащие лентивирусной трансдукции для случайной интеграции рекомбинантных TCR-кодирующих последовательностей, связанных с промотором EF1α («lenti EF1α») или промотором MND («lenti MND»), или связанных с промотором EF1α с последовательностями, кодирующими укороченный рецептора, в качестве суррогатного маркера («lenti EF1α/tReceptor»), или подвергнутые ложной трансдукции в качестве контроля («имитация») (ФИГ. 22B). На ФИГ. 22С изображен процент CD3+CD8+ клеток среди CD8+ клеток в каждой из групп, описанных выше.
[0156] На ФИГ. 23A-23C изображено связывание тетрамера пептид-MHC и поверхностная экспрессия CD8, что оценивали с помощью проточной цитометрии, для T-клеток, подвергнутых нокауту эндогенных TCR-кодирующих генов, сконструированных для экспрессии рекомбинантного T-клеточного рецептора (TCR) с использованием различных методов экспрессии : клетки, подвергали CRISPR/Cas9-опосредованному нокауту (KO) TRAC, TRBC или как TRAC, так и TRBC; клетки, подвергали направленной интеграции с помощью HDR в локусе TRAC рекомбинантных TCR-кодирующих последовательностей, связанных с промотором EF1α, промотором MND или эндогенным промотором TCR альфа, с использованием последовательности пропуска рибосомы P2A («HDR EF1α», «HDR MND» или «HDR P2A», соответственно) или клетки, подвергали ложной трансдукции в качестве контроля («ложн. трансд.») (ФИГ. 23A); клетки, сохраняющие экспрессию эндогенного TCR и подлежащие лентивирусной трансдукции для случайной интеграции рекомбинантных TCR-кодирующих последовательностей, связанных с промотором EF1α («lenti EF1α») или промотором MND («lenti MND»), или связанных с промотором EF1α с последовательностями, кодирующими укороченный рецептор в качестве суррогатного маркера («lenti EF1α/tReceptor») или подвергали ложной трансдукции в качестве контроля («ложн. трансд.») (ФИГ. 23B). На ФИГ. 23C изображен процент тетрамер+ CD8+ клеток среди CD8+ клеток в каждой из групп, описанных выше, на 7 и 13 день.
[0157] На ФИГ. 24A-24D изображена поверхностная экспрессия Vbeta22 (специфическое окрашивание для рекомбинантного TCR) и CD8, которую оценивали с помощью проточной цитометрии, для T-клеток, подвергнутых нокауту эндогенных TCR-кодирующих генов, сконструированных для экспрессии рекомбинантного T-клеточного рецептора (TCR) с использованием различных методов экспрессии: клетки подвергали CRISPR/Cas9-опосредованному нокауту (KO) TRAC, TRBC или как TRAC, так и TRBC; клетки подвергали направленной интеграции с помощью HDR в локусе TRAC рекомбинантных TCR-кодирующих последовательностей, связанных с промотором EF1α, промотором MND или эндогенным промотором TCR альфа, с использованием последовательности пропуска рибосомы P2A («HDR EF1α», «HDR MND» или «HDR P2A», соответственно), или клетки подвергали ложной трансдукции в качестве контроля («ложн. трансд.») (ФИГ. 24A); клетки, сохраняющие экспрессию эндогенного TCR и подлежащие лентивирусной трансдукции для случайной интеграции рекомбинантных TCR-кодирующих последовательностей, связанных с промотором EF1α («lenti EF1α») или промотором MND («lenti MND»), или связанных с промотором EF1α с последовательностями, кодирующими укороченный рецептор в качестве суррогатного маркера («lenti EF1α/Рецептор») или подвергали ложной трансдукции в качестве контроля («ложн. трансд.») (ФИГ. 24B). [0601] На ФИГ. 24C и 24D изображен процент Vbeta22+CD8+ клеток среди CD8+ клеток (ФИГ. 24C) и процент Vbeta22+CD4+ клеток среди CD4+ клеток (ФИГ. 24D) в каждой из групп, описанных выше, на 7 и 13 день.
[0158] На ФИГ. 25 изображена цитолитическая активность различных рекомбинантных TCR-экспрессирующих CD8+ T-клеток, как описано выше, представленная площадью под кривой (AUC) % уничтожения, по сравнению с контролем ложной трансдукции и нормализованная к экспрессии Vbeta22 для каждой группы, в результате инкубации эффекторных клеток, как описано выше, с клетками-мишенями, экспрессирующими HPV 16 Е7 при соотношении эффектора к мишени (E:T) 10:1, 5:1 и 2,5:1. CD8+ клетки, трансдуцированные лентивирусом, кодирующим эталонный TCR, способный связываться с E7 HPV 16, но содержащий мышиные области Cα и Cβ, оценивали в качестве контроля («Lenti мышь E7 эталон»).
[0159] На ФИГ. 26 изображена секреция IFNγ (пг/мл) различными рекомбинантными TCR-экспрессирующими CD8+ T-клетками, как описано выше, в результате инкубации эффекторных клеток, как описано выше, с клетками-мишенями, экспрессирующими E7 HPV 16 при соотношении эффектора к мишени (E:T) 10:1 и 2,5:1. CD8+ клетки, трансдуцированные лентивирусом, кодирующим эталонный TCR, способный связываться с E7 HPV 16, но содержащий мышиные области Cα и Cβ, оценивали в качестве контроля («Lenti мышь E7 эталон»).
[0160] На ФИГ. 27 изображена тепловая карта, показывающая относительную активность различных рекомбинантных TCR-экспрессирующих T-клеток, как описано выше, в различных функциональных анализах: AUC% уничтожения при соотношениях E:T 10:1, 5:1 и 2,5:1 («AUC»), связывание тетрамера в CD8+ клетках на 7 и 13 день («тетрамер CD8»), анализ пролиферации («подсчет CTV») с использованием клеток SCC152 или клеток-мишеней Т2, стимулированных антигенным пептидом, и секреция IFNγ из CD8+ клеток («IFNg, секретируемый CD8»).
Подробное описание изобретения
I. Т-клеточные рецепторы и другие HPV-специфичные молекулы
[0161] В настоящем описании представлены связывающие молекулы, такие как те, которые связывают или распознают пептидный эпитоп вируса папилломы человека (HPV) 16, например, пептидный эпитоп HPV 16, E6 или E7, в контексте молекулы MHC. Такие связывающие молекулы включают Т-клеточные рецепторы (TCR) и их антигенсвязывающие фрагменты и антитела и их антигенсвязывающие фрагменты, которые проявляют антигенную специфичность для связывания или распознавания пептидного эпитопа E6 HPV 16 или E7 HPV 16. Также в некоторых вариантах осуществления представлены молекулы нуклеиновых кислот, кодирующие связывающие молекулы, сконструированные клетки, содержащие связывающие молекулы, композиции и способы лечения, включающие введение таких связывающих молекул, сконструированных клеток или композиций.
[0162] В большинстве случаев HPV является возбудителем рака шейки матки и вовлечен в анальный, вагинальный, рак вульвы, полового члена и ротоглотки и другие злокачественные новообразования. Как правило, геном HPV содержит раннюю область, содержащую шесть открытых рамок считывания (E1, E2, E4, E5, E6 и E7), которые кодируют белки, участвующие в трансформации и репликации клеток, и позднюю область, содержащую две открытые рамки считывания (L1 и L2), которые кодируют белки вирусного капсида. В целом, E6 и E7 являются онкогенами, которые могут влиять на регуляцию клеточного цикла и способствовать образованию злокачественных новообразований. Например, продукт гена E6 может вызвать деградацию p53, а продукт гена E7 может вызвать инактивацию ретинобластомы (Rb).
[0163] В некоторых аспектах представленная HPV 16-связывающая молекула, включающая TCR или его антигенсвязывающий фрагмент, или антитело против HPV 16, например, фрагменты этого антитела, и белки, такие как химерные молекулы, содержащие одно или более из вышеупомянутого, такие как химерные рецепторы, например TCR-подобные CAR, и/или сконструированные клетки, экспрессирующие TCR или CAR, связываются с пептидным эпитопом, полученным из белка E6 HPV16. В некоторых аспектах представленная HPV 16-связывающая молекула, включающая TCR или его антигенсвязывающие фрагменты, или антитело против HPV 16, например фрагменты антител и содержащие их белки, такие как химерные рецепторы, например TCR-подобные CAR, и/или сконструированные клетки, экспрессирующие TCR или CAR, связываются с пептидным эпитопом, полученным из белка Е7 HPV16.
[0164] В некоторых аспектах связывающая молекула распознает или связывает эпитопы HPV 16, Е6 или Е7, в контексте молекулы МНС, такой как молекула МНС класса I. В некоторых аспектах молекула MHC класса I представляет собой молекулу HLA-A2, включая любой один или более ее подтипов, например HLA-A*0201, *0202, *0203, *0206 или *0207. В некоторых случаях могут быть различия в частоте подтипов между различными популяциями. Например, в некоторых вариантах осуществления более 95% HLA-A2-положительной европейской популяции составляют HLA-A*0201, тогда как в китайской популяции частота, как сообщается, составляет приблизительно 23% HLA-A*0201, 45% HLA-A*0207, 8% HLA-A*0206 и 23% HLA-A*0203. В некоторых вариантах осуществления молекула МНС представляет собой HLA-A*0201.
[0165] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент распознает или связывается с эпитопом или областью E6 HPV16 или E7 HPV 16, такими как пептидный эпитоп, содержащий аминокислотную последовательность, представленную в любой из SEQ ID NO: 232-239 и как показано ниже в Таблице 1.
[0166] В некоторых вариантах осуществления связывающая молекула, например TCR или его антигенсвязывающий фрагмент, или антитело или его антигенсвязывающий фрагмент, выделены или очищены или являются рекомбинантными. В некоторых аспектах связывающая молекула, например TCR или его антигенсвязывающий фрагмент, или антитело или его антигенсвязывающий фрагмент, являются человеческими. В некоторых вариантах осуществления связывающая молекула является моноклональной. В некоторых аспектах связывающая молекула представляет собой одну цепь. В других вариантах осуществления связывающая молекула содержит две цепи. В некоторых вариантах осуществления связывающая молекула, например TCR или его антигенсвязывающий фрагмент или антитело или его антигенсвязывающий фрагмент, экспрессируются на поверхности клетки.
[0167] В некоторых аспектах представленные связывающие молекулы имеют одну или более указанных функциональных особенностей, таких как свойства связывания, включая связывание с конкретными эпитопами, и/или конкретные аффинности связывания, как описано.
А. Т-клеточные рецепторы (TCR)
[0168] В некоторых вариантах осуществления связывающая молекула, которая распознает или связывает эпитоп или область HPV 16, представляет собой T-клеточный рецептор (TCR) или его антигенсвязывающий фрагмент.
[0169] В некоторых вариантах осуществления «Т-клеточный рецептор» или «TCR» представляет собой молекулу, которая содержит вариабельные α и β-цепи (также известные как TCRα и TCRβ, соответственно) или вариабельные γ- и δ-цепи (также известные как TCRγ и TCRδ, соответственно), или его антигенсвязывающие части, и которые способны специфически связываться с пептидом, связанным с молекулой МНС. В некоторых вариантах осуществления TCR представлен в форме αβ. Как правило, TCR, которые существуют в формах αβ и γδ, обычно структурно сходны, но экспрессирующие их T-клетки могут иметь различные анатомические локализации или функции. TCR может присутствовать на поверхности клетки или в растворимой форме. Обычно TCR присутствует на поверхности Т-клеток (или Т-лимфоцитов), где он обычно отвечает за распознавание антигенов, связанных с молекулами главного комплекса гистосовместимости (МНС).
[0170] Если не указано иное, термин «TCR» следует понимать как охватывающий полные TCR, а также их антигенсвязывающие части или их антигенсвязывающие фрагменты. В некоторых вариантах осуществления TCR представляет собой интактный или полноразмерный TCR, такой как TCR, содержащий α-цепь и β-цепь. В некоторых вариантах осуществления TCR представляет собой антигенсвязывающую часть, которая меньше чем полноразмерный TCR, но которая связывается со специфическим пептидом, связанным в молекуле MHC, как например, связывается с комплексом MHC-пептид. В некоторых случаях антигенсвязывающая часть или фрагмент TCR может содержать только часть структурных доменов полноразмерного или интактного TCR, но все же способна связывать пептидный эпитоп, такой как комплекс MHC-пептид, с который связывает полный TCR. В некоторых случаях антигенсвязывающая часть содержит вариабельные домены TCR, такие как вариабельная область α-цепи (Vα) и вариабельная область β-цепи (Vβ) TCR, или их антигенсвязывающие фрагменты, достаточные для образования сайта связывания для связывания с определенным комплексом MHC-пептид.
[0171] В некоторых вариантах осуществления вариабельные домены TCR содержат области, определяющие комплементарность, (CDR), которые обычно составляют основной вклад в способности распознавания и связывания антигена и специфичность пептидного, MHC и/или комплекса MHC-пептид. В некоторых вариантах осуществления CDR TCR или их комбинации образуют весь или по существу весь антигенсвязывающий сайт данной молекулы TCR. Различные CDR в вариабельной области цепи TCR обычно разделены каркасными областями (FR), которые обычно демонстрируют меньшую вариабельность среди молекул TCR по сравнению с CDR (см., например, Jores et al., Proc. Nat'l Acad. Sci. U.S.A. 87: 9138, 1990; Chothia et al., EMBO J. 7: 3745, 1988; см. также Lefranc et al., Dev. Comp. Immunol. 27:55, 2003). В некоторых вариантах осуществления CDR3 является основным CDR, ответственным за связывание или специфичность антигена, или является наиболее важным среди трех CDR в данной вариабельной области TCR для распознавания антигена и/или для взаимодействия с процессируемой пептидной частью комплекса пептид-MHC. В некоторых случаях CDR1 альфа-цепи может взаимодействовать с N-концевой частью некоторых антигенных пептидов. В некоторых контекстах CDR1 бета-цепи может взаимодействовать с С-концевой частью пептида. В некоторых контекстах CDR2 вносит наиболее сильный вклад или является основным CDR, ответственным за взаимодействие или распознавание части MHC комплекса MHC-пептид. В некоторых вариантах осуществления вариабельная область β-цепи может содержать дополнительную гипервариабельную область (CDR4 или HVR4), которая обычно участвует в связывании с суперантигеном, а не в распознавании антигена (Kotb (1995) Clinical Microbiology Reviews, 8: 411-426).
[0172] В некоторых вариантах осуществления α-цепь и/или -цепь TCR также могут содержать константный домен, трансмембранный домен и/или короткий цитоплазматический хвост (см., например, Janeway et al., Immunobiology: Immune System in Health). и Disease, 3rd Ed., Current Biology Publications, p. 4:33, 1997). В некоторых аспектах каждая цепь (например, альфа или бета) TCR может иметь один N-концевой вариабельный домен иммуноглобулина, один константный домен иммуноглобулина, трансмембранную область и короткий цитоплазматический хвост в С-концевой области. В некоторых вариантах осуществления TCR, например, через цитоплазматический хвост, ассоциирован с инвариантными белками комплекса CD3, участвующими в опосредовании сигнальной трансдукции. В некоторых случаях структура позволяет TCR связываться с другими молекулами, такими как CD3 и их субъединицы. Например, TCR, содержащий константные домены с трансмембранной областью, может закреплять белок в клеточной мембране и связываться с инвариантными субъединицами сигнального аппарата или комплекса CD3. Внутриклеточные хвосты сигнальных субъединиц CD3 (например, цепи CD3γ, CD3δ, CD3ε и CD3ζ) содержат один или более иммунорецепторных мотивов активации на основе тирозина или ITAM и обычно участвуют в сигнальной способности комплекса TCR.
[0173] Специалист в данной области может определить или идентифицировать различные домены или области TCR. В некоторых случаях точный локус домена или области может варьироваться в зависимости от конкретного структурного или гомологического моделирования или других особенностей, используемых для описания конкретного домена. Понятно, что ссылки на аминокислоты, в том числе на конкретную последовательность, представленную как SEQ ID NO, используемую для описания организации домена TCR, приведена в иллюстративных целях и не предназначена для ограничения объема представленных вариантов осуществления. В некоторых случаях конкретный домен (например, вариабельный или константный) может быть на несколько аминокислот (например, одну, две, три или четыре) длиннее или короче. В некоторых аспектах остатки TCR известны или могут быть идентифицированы в соответствии с системой нумерации Международной иммуногенетической информационной системы (IMGT) (см., например, www.imgt.org; см. также, Lefranc et al. (2003) Developmental and Comparative Immunology, 2&;55-77; and The T Cell Factsbook 2nd Edition, Lefranc and LeFranc Academic Press 2001). При использовании этой системы последовательности CDR1 в цепях Vα и/или Vβ TCR соответствуют аминокислотам, присутствующим между номерами остатков 27-38 включительно, последовательности CDR2 в цепях Vα и/или Vβ TCR соответствуют аминокислотам, присутствующим между номерами остатков 56-65 включительно, и последовательности CDR3 в цепях Vα и/или Vβ TCR соответствуют аминокислотам, присутствующим между номерами остатков 105-117 включительно.
[0174] В некоторых вариантах осуществления каждая из α-цепи и β-цепи TCR дополнительно содержат константный домен. В некоторых вариантах осуществления константный домен α-цепи (Cα) и константный домен β-цепи (Cβ) индивидуально относятся к млекопитающим, как например, константный домен человека или мыши. В некоторых вариантах осуществления константный домен соседствует с клеточной мембраной. Например, в некоторых случаях внеклеточная часть TCR, образованная двумя цепями, содержит два мембранно-проксимальных константных домена и два мембранно-дистальных вариабельных домена, каждый из которых содержит CDR.
[0175] В некоторых вариантах осуществления каждый из доменов Cα и Cβ является человеческим. В некоторых вариантах осуществления Cα кодируется геном TRAC (номенклатура IMGT) или является его вариантом. В некоторых вариантах осуществления Cα имеет или содержит последовательность аминокислот, представленную в SEQ ID NO: 213 или 220, или последовательность аминокислот, которая имеет по меньшей мере 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичности последовательности с SEQ ID NO: 213 или 220. В некоторых вариантах осуществления Cα имеет или содержит последовательность аминокислот, представленную в SEQ ID NO: 212, 215 или 217, или последовательность аминокислот, которая имеет по меньшей мере 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичности последовательности с SEQ ID NO: 212, 215 или 217. В некоторых вариантах осуществления Cα имеет или содержит последовательность аминокислот, представленную в любой из SEQ ID NO: 212, 213, 215, 217, 220 или 524. В некоторых вариантах осуществления Cβ кодируется генами TRBC1 или TRBC2 (IMGT номенклатура) или является их вариантами. В некоторых вариантах осуществления Cβ имеет или содержит последовательность аминокислот, представленную в SEQ ID NO: 214, 216, 631 или 889, или последовательность аминокислот, которая имеет по меньшей мере 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичности последовательности с SEQ ID NO: 214, 216, 631 или 889. В некоторых вариантах осуществления Cβ имеет или содержит последовательность аминокислот, представленную в SEQ ID NO: 214, 216, 631 или 889.
[0176] В некоторых вариантах осуществления любой из предложенных TCR или их антигенсвязывающих фрагментов может представлять собой химерный TCR человека/мыши. В некоторых случаях TCR или его антигенсвязывающий фрагмент содержит альфа-цепь и/или бета-цепь, содержащую константную область мыши. В некоторых вариантах осуществления Cα представляет собой константную область мыши, которая представляет собой или содержит последовательность аминокислот, представленную в SEQ ID NO: 262, 317, 833, 1012, 1014, 1015, 1017 или 1018, или последовательность аминокислот, которая демонстрирует по меньшей мере 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичности последовательности с SEQ ID NO: 262, 317, 833, 1012, 1014, 1015, 1017 или 1018. В некоторых вариантах осуществления Cα представляет собой или содержит последовательность аминокислот, представленную в SEQ ID NO: 262, 317, 833, 1012, 1014, 1015, 1017 или 1018. В некоторых вариантах осуществления Cβ представляет собой константную область мыши, которая представляет собой или содержит последовательность аминокислот, представленную в SEQ ID NO: 263, 109, 1013 или 1016, или последовательность аминокислот, которая демонстрирует по меньшей мере 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичности последовательности с SEQ ID NO: 263, 109 1013 или 1016. В некоторых вариантах осуществления Cβ представляет собой или содержит последовательность аминокислот, представленную в SEQ ID NO: 263, 109, 1013 или 1016. В некоторых вариантах осуществления Cα представляет собой или содержит последовательность аминокислот, представленную в SEQ ID NO: 262 или 1014, или последовательность аминокислот, которая имеет по меньшей мере 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичности последовательности с SEQ ID NO: 262 или 1014, и/или Cβ представляет собой или содержит последовательность аминокислот, представленную в SEQ ID NO: 263, или последовательность аминокислот, которая демонстрирует по меньшей мере 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94% 95%, 96%, 97%, 98%, 99% или более идентичности последовательности с SEQ ID NO: 263. В некоторых вариантах осуществления Cα и/или Cβ представляет собой или содержит любые Cα и/или Cβ, описанные в WO 2015/184228, WO 2015/009604 и WO 2015/009606.
[0177] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент в данном документе содержит вариант альфа-цепи и/или бета-цепи, например альфа- и/или бета-цепи, которая содержит константную область мыши. В некоторых вариантах осуществления этот вариант содержит аминокислотную последовательность любого из TCR, описанных в настоящем документе, с одной, двумя, тремя или четырьмя или более аминокислотными заменами в константной области альфа или бета-цепи. В некоторых вариантах осуществления вариант содержит аминокислотную последовательность любой из константных областей, описанных в настоящем документе, с одной, двумя, тремя или четырьмя или более аминокислотными заменами в константной области. В некоторых вариантах осуществления TCR (или их функциональные части), содержащие замещенную аминокислотную последовательность(и), преимущественно обеспечивают одно или более из улучшенного распознавания мишеней HPV 16, повышенной экспрессии клеткой-хозяином и повышенной противоопухолевой активности по сравнению с родительским TCR, содержащим незамещенную аминокислотную последовательность.
[0178] В некоторых вариантах осуществления замещенные аминокислотные последовательности константных областей мыши α и β-цепей TCR, SEQ ID NO: 1015 и 1016, соответственно, соответствуют всем или частям незамещенных аминокислотных последовательностей константной области мыши SEQ ID NO: 1014 и 263, соответственно, с SEQ ID NO: 1015, имеющей одну, две, три или четыре аминокислотные замены по сравнению с SEQ ID NO: 1014 и SEQ ID NO: 1016, имеющими одну аминокислотную замену по сравнению с SEQ ID NO: 263. В некоторых вариантах осуществления вариант TCR содержит аминокислотные последовательности (а) SEQ ID NO: 1015 (константная область альфа-цепи), где (i) X в положении 48 представляет собой Thr или Cys; (ii) X в положении 112 представляет собой Ser, Gly, Ala, Val, Leu, Ile, Pro, Phe, Met или Trp; (iii) X в положении 114 представляет собой Met, Gly, Ala, Val, Leu, Ile, Pro, Phe, Met или Trp; и (iv) X в положении 115 представляет собой Gly, Ala, Val, Leu, Ile, Pro, Phe, Met или Trp; и (b) SEQ ID NO: 1016 (константная область бета-цепи), где X в положении 56 представляет собой Ser или Cys. В некоторых вариантах осуществления Cα представляет собой или содержит последовательность аминокислот, представленную в SEQ ID NO: 1015, или последовательность аминокислот, которая имеет по меньшей мере 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичности последовательности с SEQ ID NO: 1015, и/или Cβ представляет собой или содержит последовательность аминокислот, представленную в SEQ ID NO: 1016 или последовательность аминокислот, которая имеет по меньшей мере 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичности последовательности с SEQ ID NO: 1016.
[0179] В некоторых вариантах осуществления TCR может представлять собой гетеродимер из двух цепей α и β, которые связаны, например, посредством дисульфидной связи или дисульфидных связей. В некоторых вариантах осуществления константный домен TCR может содержать короткие соединительные последовательности, в которых остаток цистеина образует дисульфидную связь, тем самым связывая две цепи TCR. В некоторых вариантах осуществления TCR может иметь дополнительный остаток цистеина в каждой из α и β цепей, так что TCR содержит две дисульфидные связи в константных доменах. В некоторых вариантах осуществления каждый из константного и вариабельного доменов содержит дисульфидные связи, образованные остатками цистеина.
[0180] В некоторых вариантах осуществления TCR может содержать введенную дисульфидную связь или связи. В некоторых вариантах осуществления нативные дисульфидные связи отсутствуют. В некоторых вариантах осуществления один или более нативных цистеинов (например, в константном домене α-цепи и β-цепи), которые образуют природную межцепочечную дисульфидную связь, замещены другим остатком, таким как серин или аланин. В некоторых вариантах осуществления введенная дисульфидная связь может быть образована путем мутирования нецистеиновых остатков на альфа- и бета-цепях, как например, в константном домене α-цепи и β-цепи, на цистеин. Противоположные цистеины в α- и β-цепях TCR обеспечивают дисульфидную связь, которая связывает константные области α- и β-цепей TCR замещенного TCR друг с другом и которая отсутствует в TCR, содержащем незамещенную константную область человека или незамещенную константную область мыши. В некоторых вариантах осуществления присутствие ненативных остатков цистеина (например, приводящих к одной или более ненативным дисульфидным связям) в рекомбинантном TCR может способствовать продуцированию желаемого рекомбинантного TCR в клетке, в которую он введен, сверхэкспрессии несоответствующей пары TCR, содержащей нативную цепь TCR.
[0181] Иллюстративные ненативные дисульфидные связи TCR описаны в опубликованных Международных РСТ № WO 2006/000830 и WO 2006/037960. В некоторых вариантах осуществления цистеины могут быть введены или замещены в остатке Thr48 цепи Cα и Ser57 цепи Cβ, в остатке Thr45 цепи Cα и Ser77 цепи Cβ, в остатке Tyr10 цепи Cα и Ser17 в Cβ цепи, в остатке Thr45 цепи Cα и Asp59 цепи Cβ и/или в остатке Ser15 цепи Cα и Glu15 цепи Cβ относительно нумерации Cα, представленной в любой из SEQ ID NO: 212, 213, 217 или 524, или Cβ, представленной в SEQ ID NO: 214 или 216. В некоторых вариантах осуществления вариант TCR представляет собой цистеин-замещенный химерный TCR, в котором один или оба из нативного Thr48 из SEQ ID NO: 1014 и нативного Ser57 из SEQ ID NO: 263 замещены Cys. В некоторых вариантах осуществления Cα представляет собой или содержит последовательность аминокислот, представленную в SEQ ID NO: 1017, или последовательность аминокислот, которая имеет по меньшей мере 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичности последовательности с SEQ ID NO: 1017, и/или Cβ представляет собой или содержит последовательность аминокислот, представленную в SEQ ID NO: 1016, или последовательность аминокислот, которая имеет по меньшей мере 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичности последовательности с SEQ ID NO: 1013.
[0182] В некоторых вариантах осуществления любая из представленных мутаций цистеина может быть осуществлена в соответствующем положении в другой последовательности, например, в последовательностях мышиных Cα и Cβ, описанных выше. Термин «соответствующий» по отношению к положениям белка, как например, указание, что аминокислотные положения «соответствуют» аминокислотным положениям в раскрытой последовательности, такой как представлено в списке последовательностей, относится к аминокислотным положениям, идентифицированным после выравнивания с раскрытой последовательностью на основе структурного выравнивания последовательностей или с использованием стандартного алгоритма выравнивания, такого как алгоритм GAP. Например, соответствующие остатки могут быть определены путем выравнивания эталонной последовательности с последовательностью Cα, представленной в любой из SEQ ID NO: 212, 213, 215, 217, 220 или 524, или последовательностью Cβ, представленной в SEQ ID NO: 214, 216, 631 или 889, методами структурного выравнивания, как описано в настоящем документе. Выравнивая последовательности, специалист в данной области может идентифицировать соответствующие остатки, например, используя консервативные и идентичные аминокислотные остатки в качестве направляющих.
[0183] В некоторых вариантах осуществления этот вариант включает замены одной, двух или трех аминокислот в трансмембранном (ТМ) домене константной области одной или обеих α- и β-цепей гидрофобной аминокислотой для получения TCR с замещенной гидрофобной аминокислотой. Гидрофобная аминокислотная замена(замены) в домене ТМ TCR может увеличить гидрофобность домена ТМ TCR по сравнению с TCR, в котором отсутствует гидрофобная аминокислотная замена(замены) в домене ТМ. В некоторых вариантах осуществления вариант TCR содержит один, два или три нативных Ser 112, Met 114 и Gly 115 SEQ ID NO: 1014, и может быть независимо замещен с помощью Gly, Ala, Val, Leu, Ile, Pro, Phe, Met или Trp; например, с Leu, Ile или Val. В некоторых вариантах осуществления Cα представляет собой или содержит последовательность аминокислот, представленную в SEQ ID NO: 1018, или последовательность аминокислот, которая имеет по меньшей мере 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичности последовательности с SEQ ID NO: 1018, и/или Cβ представляет собой или содержит последовательность аминокислот, представленную в SEQ ID NO: 263, или последовательность аминокислот, которая имеет по меньшей мере 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичности последовательности с SEQ ID NO: 263.
[0184] В некоторых вариантах осуществления этот вариант включает замены цистеина в константной области одной или обеих α- и β-цепей в комбинации с замещением(замещениями) одной, двух или трех аминокислот в трансмембранном (TM) домене константной области одной или обеих α- и β-цепей на гидрофобную аминокислоту. В некоторых вариантах осуществления вариант имеет нативный Thr48 SEQ ID NO: 1014, замещенный на Cys; один, два или три из нативного Ser 112, Met 114 и Gly 115 SEQ ID NO: 1014, независимо, замещенных на Gly, Ala, Val, Leu, Ile, Pro, Phe, Met или Trp; например, на Leu, Ile или Val; и нативный Ser56 SEQ ID NO: 19, замещенный на Cys. В некоторых вариантах осуществления Cα представляет собой или содержит последовательность аминокислот, представленную в SEQ ID NO: 833, или последовательность аминокислот, которая имеет по меньшей мере 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичности последовательности с SEQ ID NO: 833, и/или Cβ представляет собой или содержит последовательность аминокислот, представленную в SEQ ID NO: 1013Ю, или последовательность аминокислот, которая имеет по меньшей мере 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичности последовательности с SEQ ID NO: 1013.
[0185] Иллюстративные последовательности (например, CDR, Vα и/или последовательности константной области Vβ) предложенных TCR описаны ниже.
[0186] В некоторых вариантах осуществления TCR является полноразмерным TCR. В некоторых вариантах осуществления TCR представляет собой антигенсвязывающую часть. В некоторых вариантах осуществления TCR представляет собой димерный TCR (dTCR). В некоторых вариантах осуществления TCR представляет собой одноцепочечный TCR (sc-TCR). TCR может быть связанным с клеткой или в растворимой форме. В некоторых вариантах осуществления TCR находится в связанной с клеткой форме, экспрессируемой на поверхности клетки.
[0187] В некоторых вариантах осуществления dTCR содержит первый полипептид, где последовательность, соответствующая представленной последовательности вариабельной области α-цепи TCR, слита с N-концом последовательности, соответствующей внеклеточной последовательности константной области α-цепи TCR, и второй полипептид, где последовательность, соответствующая представленной последовательности вариабельной области β-цепи TCR, слита с N-концом последовательности, соответствующей внеклеточной последовательности константной области β-цепи TCR, причем первый и второй полипептиды связаны дисульфидной связью. В некоторых вариантах осуществления связь может соответствовать нативной межцепочечной дисульфидной связи, присутствующей в нативных димерных αβ TCR. В некоторых вариантах осуществления межцепные дисульфидные связи отсутствуют в нативном TCR. Например, в некоторых вариантах осуществления один или более цистеинов могут быть включены во внеклеточные последовательности константной области полипептидной пары dTCR. В некоторых случаях может быть желательной как нативная, так и ненативная дисульфидная связь. В некоторых вариантах осуществления TCR содержит трансмембранную последовательность для закрепления на мембране.
[0188] В некоторых вариантах осуществления dTCR содержит представленную α-цепь TCR, содержащую вариабельный α-домен, константный α-домен и первый мотив димеризации, присоединенный к C-концу константного α-домена, и представленную β-цепь TCR, содержащую вариабельный β-домен, константный β-домен и первый мотив димеризации, присоединенный к C-концу константного β-домена, где первый и второй мотивы димеризации легко взаимодействуют, образуя ковалентную связь между аминокислотой в первом мотиве димеризации и аминокислотой в втором мотиве димеризации, связывая α-цепь TCR и β-цепь TCR вместе.
[0189] В некоторых вариантах осуществления TCR представляет собой scTCR, который представляет собой одиночную аминокислотную цепь, содержащую α-цепь и β-цепь, который способен связываться с комплексами MHC-пептид. Как правило, scTCR можно генерировать с использованием способов, известных специалистам в данной области техники, см., например, Международные опубликованные PCT No. WO 96/13593, WO 96/18105, WO 99/18129, WO 04/033685, WO 2006/037960, WO 2011/044186; Патент США № 7569664; и Schlueter, C.J. et al. J. Mol. Biol. 256, 859 (1996).
[0190] В некоторых вариантах осуществления scTCR содержит первый сегмент, состоящий из аминокислотной последовательности, соответствующей последовательности представленной вариабельной области α-цепи TCR, второй сегмент, состоящий из аминокислотной последовательности, соответствующей представленной последовательности вариабельной области β-цепи TCR, слитой с N-концом аминокислотной последовательности, соответствующей внеклеточной последовательности константного домена β-цепи TCR, и линкерную последовательность, связывающую C-конец первого сегмента с N-концом второго сегмента.
[0191] В некоторых вариантах осуществления scTCR содержит первый сегмент, состоящий из аминокислотной последовательности, соответствующей представленной вариабельной области β-цепи TCR, второй сегмент, состоящий из аминокислотной последовательности, соответствующей представленной последовательности вариабельной области α-цепи TCR, слитой с N-концом аминокислотной последовательности, соответствующей внеклеточной последовательности константного домена α-цепи TCR, и линкерную последовательность, связывающую C-конец первого сегмента с N-концом второго сегмента.
[0192] В некоторых вариантах осуществления scTCR содержит первый сегмент, состоящий из представленной последовательности вариабельной области α-цепи, слитой с N-концом последовательности внеклеточного константного домена α-цепи, и второй сегмент, состоящий из представленной последовательности вариабельной области β-цепи, слитой с N концом последовательности внеклеточного константного домена β-цепи, и трансмембранную последовательность и, необязательно, линкерную последовательность, связывающую C-конец первого сегмента с N-концом второго сегмента.
[0193] В некоторых вариантах осуществления scTCR содержит первый сегмент, состоящий из представленной последовательности вариабельной области β-цепи TCR, слитой с N-концом последовательности внеклеточного константного домена β-цепи, и второй сегмент, состоящий из представленной последовательности вариабельной области α-цепи, слитой с N-концом последовательности внеклеточного константного домена α-цепи, и трансмембранную последовательность и, необязательно, линкерную последовательность, связывающую C-конец первого сегмента с N-концом второго сегмента.
[0194] В некоторых вариантах осуществления, чтобы scTCR связывался с комплексом MHC-пептид, α- и β-цепи должны быть спарены так, чтобы их последовательности вариабельных областей были ориентированы для такого связывания. Различные способы стимулирования спаривания α и β в scTCR хорошо известны в данной области. В некоторых вариантах осуществления включена линкерная последовательность, которая связывает α- и β-цепи с образованием единой полипептидной цепи. В некоторых вариантах осуществления линкер должен иметь достаточную длину, чтобы охватить расстояние между C-концом α-цепи и N-концом β-цепи или наоборот, при этом также гарантируя, что длина линкера не будет такой длинной, чтобы он блокировал или уменьшал связывание scTCR с целевым комплексом пептид-МНС.
[0195] В некоторых вариантах осуществления линкер scTCR, который связывает первый и второй сегменты TCR, может представлять собой любой линкер, способный образовывать одну цепь полипептида, сохраняя при этом специфичность связывания TCR. В некоторых вариантах осуществления линкерная последовательность может, например, иметь формулу -P-AA-P-, где P представляет собой пролин, а AA представляет собой аминокислотную последовательность, где аминокислотами являются глицин и серин. В некоторых вариантах осуществления первый и второй сегменты спарены так, что их последовательности вариабельных областей ориентированы для такого связывания. Следовательно, в некоторых случаях линкер имеет достаточную длину, чтобы охватить расстояние между C-концом первого сегмента и N-концом второго сегмента или наоборот, но пр этом он не слишком длинный, чтобы блокировать или уменьшать связывание scTCR с целевым лигандом. В некоторых вариантах осуществления линкер может содержать или может примерно содержать 10-45 аминокислот, как например, 10-30 аминокислот или 26-41 аминокислотный остаток, например, 29, 30, 31 или 32 аминокислоты. В некоторых вариантах осуществления линкер имеет формулу -PGGG- (SGGGG) n-P-, где n равно 5 или 6, и P представляет собой пролин, G представляет собой глицин, и S представляет собой серин (SEQ ID NO: 266). В некоторых вариантах осуществления линкер имеет последовательность GSADDAKKDAAKKDGKS (SEQ ID NO: 267).
[0196] В некоторых вариантах осуществления scTCR содержит дисульфидную связь между остатками одной аминокислотной цепи, которая в некоторых случаях может способствовать стабильности спаривания между α- и β-областями одноцепочечной молекулы (см., например, Патент США No. 7569664). В некоторых вариантах осуществления scTCR содержит ковалентную дисульфидную связь, связывающую остаток иммуноглобулиновой области константного домена α-цепи с остатком иммуноглобулиновой области константного домена β-цепи одноцепочечной молекулы. В некоторых вариантах осуществления дисульфидная связь соответствует нативной дисульфидной связи, присутствующей в нативном dTCR. В некоторых вариантах осуществления дисульфидная связь в нативном TCR отсутствует. В некоторых вариантах осуществления дисульфидная связь представляет собой введенную ненативную дисульфидную связь, например, путем включения одного или более цистеинов во внеклеточные последовательности константной области областей первой и второй цепей полипептида scTCR. Иллюстративные мутации цистеина включают любые, как описано выше. В некоторых случаях может присутствовать как нативная, так и ненативная дисульфидная связь.
[0197] В некоторых вариантах осуществления scTCR представляет собой укороченный TCR, связанный недисульфидной связью, в котором гетерологичные лейциновые застежки, слитые с их С-концами, облегчают ассоциацию цепи (см., например, международную публикацию PCT No. WO99/60120). В некоторых вариантах осуществления scTCR содержит вариабельный домен TCRα, ковалентно связанный с вариабельным доменом TCRβ через пептидный линкер (см., например, международную публикацию PCT No. WO99/18129).
[0198] В некоторых вариантах осуществления любой из представленных TCR, включая dTCR или scTCR, может быть связан с сигнальными доменами, которые способствуют получению активного TCR на поверхности T-клетки. В некоторых вариантах осуществления TCR экспрессируется на поверхности клеток. В некоторых вариантах осуществления TCR содержит последовательность, соответствующую трансмембранной последовательности. В некоторых вариантах осуществления трансмембранный домен является положительно заряженным. В некоторых вариантах осуществления трансмембранный домен может представлять собой трансмембранный домен Cα или Cβ. В некоторых вариантах осуществления трансмембранный домен может происходить из источника, отличного от TCR, например, из трансмембранной области CD3z, CD28 или B7.1. В некоторых вариантах осуществления TCR содержит последовательность, соответствующую цитоплазматическим последовательностям. В некоторых вариантах осуществления TCR содержит сигнальный домен CD3z. В некоторых вариантах осуществления TCR способен образовывать комплекс TCR с CD3.
[0199] В некоторых вариантах осуществления TCR представляет собой растворимый TCR. В некоторых вариантах осуществления растворимый TCR имеет структуру, как описано в WO 99/60120 или WO 03/020763. В некоторых вариантах осуществления TCR не содержит последовательности, соответствующей трансмембранной последовательности, например, чтобы позволить мембранное закрепление в клетке, в которой он экспрессируется. В некоторых вариантах осуществления TCR не содержит последовательности, соответствующей цитоплазматическим последовательностям.
Иллюстративные TCR
[0200] В некоторых вариантах осуществления среди представленных TCR или их антигенсвязывающих фрагментов, которые связывают или распознают пептидный эпитоп HPV 16 в контексте MHC (например, пептидный эпитоп E6 HPV 16 или пептидный эпитоп E7 HPV 16), присутствуют TCR или их антигенсвязывающие фрагменты, которые содержат любую из последовательностей вариабельной области альфа- и/или бета-цепи (Vα или Vβ), как описано, индивидуально, или достаточную антигенсвязывающую часть такой цепи(цепей). В некоторых вариантах осуществления представленный анти-HPV 16 TCRили его антигенсвязывающий фрагмент (например, анти-E6 HPV 16 или анти-E7 HPV 16 TCR) содержит последовательность области Vα или ее достаточную антигенсвязывающую часть, которая содержит CDR-1, CDR-2 и/или CDR-3, как описано. В некоторых вариантах осуществления представленный анти-HPV 16 TCR или его антигенсвязывающий фрагмент (например, анти-E6 HPV 16 или анти-E7 HPV 16 TCR) содержит последовательность области Vβ или достаточную антигенсвязывающую часть, которая содержит CDR-1, CDR-2 и/или CDR-3, как описано. В некоторых вариантах осуществления анти-HPV 16 TCR или его антигенсвязывающий фрагмент (например, анти-E6 HPV 16 или анти-E7 HPV 16 TCR) содержат последовательность области Vα, которая содержит CDR-1, CDR-2 и/или CDR-3, как описано, и содержит последовательность области Vβ, которая содержит CDR-1, CDR-2 и/или CDR-3, как описано. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере на или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99%, идентичны такой последовательности.
[0201] В некоторых вариантах осуществления TCR содержит область Vα, которая содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность X1X2X3X4X5X6X7X8X9X10X11X12X13X14X15X16X17X18 (SEQ ID NO: 1365), где X1 представляет собой A, I или V; Х2 представляет собой М, L, V, Е или А; X3 представляет собой R, L, N или S; X4 представляет собой E, V, P, T, F, I, R или A; X5 представляет собой G, I, L, A, P, R, D или H; X6 представляет собой R, T, G, S, N или H; X7 представляет собой G, R, A, N или отсутствует; X8 представляет собой T, G или отсутствует; X9 отсутствует или представляет собой A или G; Х10 отсутствует или представляет собой G; X11 отсутствует или представляет собой G; X12 отсутствует или представляет собой T; X13 представляет собой F, Y, A, S или отсутствует; X14 представляет собой G, Y или N; X15 представляет собой F, G, T, N, Q или Y; X16 представляет собой K, P, V, N или A; X17 представляет собой T, L или F; и X18 представляет собой I, V, T, H или N.
[0202] В некоторых вариантах осуществления TCR содержит область Vα, которая содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность X1X2X3X4X5X6X7X8X9X10X11X12X13X14X15X16X17X18 (SEQ ID NO: 251), где X1 представляет собой A, I или V; Х2 представляет собой М, L, V, E или S; X3 представляет собой R, L, N, Q, P или S; X4 представляет собой E, V, P, T, F, I, R, G, S или A; X5 представляет собой G, I, L, A, P, R, D, отсутствует или H; X6 представляет собой R, T, G, S, N, отсутствует или представляет собой А; X7 представляет собой G, R, N или отсутствует; X8 представляет собой T, G или отсутствует; X9 отсутствует или представляет собой A; Х10 отсутствует или представляет собой G; X11 отсутствует или представляет собой G; X12 отсутствует или представляет собой T; X13 представляет собой F, Y, S или отсутствует; X14 представляет собой G, Y, отсутствует или представляет собой N; X15 представляет собой F, G, T, N, Q или Y; X16 представляет собой K, P, V, N или A; X17 представляет собой T, L или F; и X18 представляет собой I, V, T, H, F или N.
[0203] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержит область Vα, содержащую область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 138, 144, 147, 153, 159, 163, 167, 173, 175, 301, 304, 308, 478, 493, 505, 511, 523, 539, 555, 572, 588, 600, 612, 624, 638, 650, 662, 679, 694, 712, 729, 744, 762, 776, 788, 802, 818, 832, 846, 858, 870, 882, 896, 911, 926, 940, 952, 964, 976, 988 или 1002, или последовательность, имеющую по меньшей мере или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью. В некоторых аспектах TCR или его антигенсвязывающий фрагмент содержит область Vα, содержащую CDR3, содержащуюся в аминокислотной последовательности, представленной в любой из SEQ ID NO: 111, 113, 115, 117, 119, 121, 123, 125, 127, 295, 297, 299, 477, 492, 504, 510, 522, 536, 554, 569, 587, 599, 611, 623, 637, 649, 661, 676, 691, 709, 726, 741, 759, 775, 787, 799, 815, 830, 845, 857, 869, 881, 895, 908, 925, 937, 951, 963, 975, 987 или 999, или последовательности, которые по меньшей мере, или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью.
[0204] В некоторых вариантах осуществления TCR содержит область Vβ, которая содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность X1X2X3X4X5X6X7X8X9X10X11X12X13X14X15 (SEQ ID NO: 1366), где X1 представляет собой A или S; Х2 представляет собой S, I или V; X3 представляет собой S, T или V; X4 представляет собой H, P, L, Y, T, D или Q; Х5 представляет собой L, G, W, F, S или R; X6 представляет собой A, G, L, S или T; X7 представляет собой G, E, A, T, R или отсутствует; Х8 отсутствует или представляет собой G; X9 отсутствует или представляет собой G; X10 отсутствует, F, G, T, S или A; X11 представляет собой T, N, H, A, S или F; X12 представляет собой G, T, Q, D, Y или L; X13 представляет собой E, P, T, G или W; X14 представляет собой L, A, Q, Y или K; и X15 представляет собой F, H, Y или T.
[0205] В некоторых вариантах осуществления TCR содержит область Vβ, которая содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность X1X2X3X4X5X6X7X8X9X10X11X12X13X14X15 (SEQ ID NO: 261), где X1 представляет собой A или S; Х2 представляет собой S, I или V; X3 представляет собой S, T или V; X4 представляет собой H, P, L, Y, T, D или F; Х5 представляет собой L, G, W, F, S, T или R; X6 представляет собой A, G, L, S или T; X7 представляет собой G, E, A, T, R, Q или отсутствует; Х8 отсутствует или представляет собой G; X9 отсутствует или представляет собой G; X10 отсутствует, F, G, T, S или R; X11 представляет собой T, N, H, A, S, R или E; X12 представляет собой G, T, Q, D, Y или R; X13 представляет собой E, P, T или G; X14 представляет собой L, A, Q или Y; и X15 представляет собой F, H, Y или T.
[0206] В некоторых случаях TCR содержит область Vβ, содержащую область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 141, 146, 150, 156, 160, 164, 170, 174 178, 305, 309, 486, 499, 517, 531, 548, 563, 581, 594, 606, 618, 630, 644, 656, 670, 686, 703, 721, 736, 753, 769, 782, 794, 809, 825, 840, 852, 864, 876, 888, 902, 919, 932, 946, 958, 970, 982, 994, 1010 или 1381, или последовательность, имеющую по меньшей мере или примерно 90 или 91, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью. В некоторых вариантах осуществления TCR содержит область Vβ, содержащую CDR3, содержащуюся в аминокислотной последовательности, представленной в любой из SEQ ID NO: 112, 114, 116, 118, 120, 122, 124, 126, 128, 296, 298, 300, 483, 498, 516, 530, 545, 560, 578, 593, 605, 617, 629, 643, 655, 667, 685, 700, 718, 735, 750, 768, 781, 793, 808, 824, 839, 851, 863, 875, 887, 901, 917, 931, 945, 957, 969, 981, 993, 1008 или 1380, или последовательности, которая по меньшей мере, или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентична такой последовательности.
[0207] В некоторых аспектах область Vα дополнительно содержит область 1, определяющую комплементарность, (CDR-1), содержащую аминокислотную последовательность X1X2X3X4X5X6X7 (SEQ ID NO: 1367), где X1 представляет собой T, D, N или V; Х2 представляет собой I или S; X3 представляет собой S, D, A, P или M; X4 представляет собой G, Q, P или отсутствует; X5 представляет собой T, S, I или F; X6 представляет собой D, Y, Q, T или S; и X7 представляет собой Y, G, N или Q. В некоторых вариантах осуществления область Vα дополнительно содержит область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность X1X2X3X4X5X6X7X8 (SEQ ID NO: 247), где X1 представляет собой G, Q, I, V или M; X2 представляет собой L, S, Q, Y, F, T или G; X3 представляет собой T, G, S или F; X4 представляет собой Y, S, N, I или отсутствует; Х5 отсутствует или представляет собой D; X6 отсутствует, E, Q, S, M или K; X7 представляет собой S, Q, R, G, D или N; и X8 представляет собой N, E, M, T или K.
[0208] В некоторых аспектах область Vα дополнительно содержит область 1, определяющую комплементарность, (CDR-1), содержащую аминокислотную последовательность X1X2X3X4X5X6X7 (SEQ ID NO: 243), где X1 представляет собой T, D, N, S или V; Х2 представляет собой I или S; X3 представляет собой S, D, A, P, N или Y; X4 представляет собой G, Q, P или отсутствует; X5 представляет собой T, S, I или F; X6 представляет собой D, Y, Q, T, P или S; и X7 представляет собой Y, G, N, A, S или Q.
[0209] В некоторых вариантах осуществления область Vα содержит область 1, определяющую комплементарность, (CDR-1), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 136, 142, 151, 157, 161, 165, 171, 302, 306, 537, 570, 677, 692, 710, 727, 742, 760, 800, 816, 909, 938 или 1000, или последовательность, имеющую по меньшей мере или примерно 90 или 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью. В некоторых аспектах область Vα содержит CDR-1, содержащуюся в аминокислотной последовательности, представленной в любой из SEQ ID NO: 111, 113, 115, 117, 119, 121, 123, 125, 127, 295, 297, 299. 477, 492, 504, 510, 522, 536, 554, 569, 587, 599, 611, 623, 637, 649, 661, 676, 691, 709, 726, 741, 759, 775, 787, 799, 815, 830, 845, 857, 869, 881, 895, 908, 925, 937, 951, 963, 975, 987 или 999 или последовательности, имеющей по меньшей мере или примерно 90 или 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью. В некоторых вариантах осуществления область Vα содержит область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 137, 143, 152, 158, 162, 166, 172, 303, 307, 538, 571, 678, 693, 711, 728, 743, 761, 801, 817, 831, 910, 939 или 1001, или последовательность, имеющую по меньшей мере или примерно 90 или 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью. В некоторых вариантах осуществления область Vα содержит CDR-2, содержащуюся в аминокислотной последовательности, представленной в любой из SEQ ID NO: 111, 113, 115, 117, 119, 121, 123, 125, 127, 295, 297, 299. 477, 492, 504, 510, 522, 536, 554, 569, 587, 599, 611, 623, 637, 649, 661, 676, 691, 709, 726, 741, 759, 775, 787, 799, 815, 830, 845, 857, 869, 881, 895, 908, 925, 937, 951, 963, 975, 987 или 999, или последовательности, имеющей по меньшей мере или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью.
[0210] В некоторых аспектах область Vβ дополнительно содержит область 1, определяющую комплементарность, (CDR-1), содержащую аминокислотную последовательность X1X2X3X4X5 (SEQ ID NO: 1369), где X1 представляет собой S, M или L; X2 представляет собой G, E, D, N или Q; Х3 представляет собой Н или V; X4 представляет собой V, N, E, L или T; и X5 представляет собой S, R, N, Y, A или M. В некоторых вариантах осуществления область Vβ дополнительно содержит область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность X1X2X3X4X5X6X7 (SEQ ID NO: 1368), где X1 представляет собой F, Y, S или A; Х2 представляет собой Q, Y, V или N; X3 представляет собой N, D, G, F или Q; X4 отсутствует или представляет собой G; X5 представляет собой E, V, N, K или S; X6 представляет собой A, K, G или E; и X7 представляет собой Q, M, T, I или A.
[0211] В некоторых аспектах область Vβ дополнительно содержит область 1, определяющую комплементарность, (CDR-1), содержащую аминокислотную последовательность X1X2X3X4X5 (SEQ ID NO: 254), где X1 представляет собой S или M; X2 представляет собой G, E, D, N или Q; Х3 представляет собой Н или V; X4 представляет собой V, N, E, L или T; и X5 представляет собой S, R, N, Y или M. В некоторых вариантах осуществления область Vβ дополнительно содержит область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность X1X2X3GX5X6X7 (SEQ ID NO: 257), где X1 представляет собой F, S или A; Х2 представляет собой Q, Y, V или N; X3 представляет собой N, D, G или Q; Х5 представляет собой Е, V, N или S; X6 представляет собой A, K, G или E; и X7 представляет собой Q, M, T, I или A.
[0212] В некоторых случаях область Vβ содержит область 1, определяющую комплементарность, (CDR-1), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 139, 145, 148, 154, 168, 176, 484, 546, 561, 579, 668, 701, 719 или 751, или последовательность, имеющую, по меньшей мере или примерно 90 или 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью. В некоторых аспектах область Vβ содержит CDR-1, содержащуюся в аминокислотной последовательности, представленной в любой из SEQ ID NO: 112, 114, 116, 118, 120, 122, 124, 126, 128, 296, 298, 300. 483, 498, 516, 530, 545, 560, 578, 593, 605, 617, 629, 643, 655, 667, 685, 700, 718, 735, 750, 768, 781, 793, 808, 824, 839, 851, 863, 875, 887, 901, 917, 931, 945, 957, 969, 981, 993, 1008 или 1380, или последовательности, имеющей по меньшей мере или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью. В некоторых вариантах осуществления область Vβ содержит область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 140, 149, 155, 169, 177, 485, 547, 562, 580, 669, 702, 720, 752, 918 или 1009, или последовательность, имеющую, по меньшей мере или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью. В некоторых вариантах осуществления область Vβ содержит CDR-2, содержащуюся в аминокислотной последовательности, представленной в любой из SEQ ID NO: 112, 114, 116, 118, 120, 122, 124, 126, 128, 296, 298, 300. 483, 498, 516, 530, 545, 560, 578, 593, 605, 617, 629, 643, 655, 667, 685, 700, 718, 735, 750, 768, 781, 793, 808, 824, 839, 851, 863, 875, 887, 901, 917, 931, 945, 957, 969, 981, 993, 1008 или 1380, или последовательности, имеющей по меньшей мере или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью.
[0213] В некоторых вариантах осуществления область Vα содержит аминокислотную последовательность, представленную в любой из SEQ ID NO: 111, 113, 115, 117, 119, 121, 123, 125, 127, 295, 297, 299, 477, 492, 504. 510, 522, 536, 554, 569, 587, 599, 611, 623, 637, 649, 661, 676, 691, 709, 726, 741, 759, 775, 787, 799, 815, 830, 845, 857, 869, 881, 895, 908, 925, 937, 951, 963, 975, 987 или 999, или аминокислотную последовательность, которая имеет с ними по меньшей мере 90%, 91%, 92%, 93%, 94%, 95% 96%, 97%, 98% или 99% идентичности последовательности. В некоторых случаях область Vβ содержит аминокислотную последовательность, представленную в любой из SEQ ID NO: 112, 114, 116, 118, 120, 122, 124, 126, 128, 296, 298, 300, 483, 498, 516. 530, 545, 560, 578, 593, 605, 617, 629, 643, 655, 667, 685, 700, 718, 735, 750, 768, 781, 793, 808, 824, 839, 851, 863, 875, 887, 901, 917, 931, 945, 957, 969, 981, 993 или 1008, или аминокислотную последовательность, которая имеет с ними по меньшей мере 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% идентичности последовательности. В некоторых вариантах осуществления TCR содержит альфа-цепь, содержащую любую из таких последовательностей Vα-цепи и любую из таких последовательностей Vβ-цепи.
[0214] В некоторых вариантах осуществления альфа-цепь TCR или его антигенсвязывающего фрагмента дополнительно содержит константную область альфа (Cα) или ее часть. В некоторых аспектах бета-цепь дополнительно содержит константную область бета (Cβ) или ее часть. Таким образом, в некоторых вариантах осуществления TCR, например, E6 или E7 HPV 16 TCR или его антигенсвязывающий фрагмент содержит альфа-цепь, содержащую вариабельную область альфа (Vα) и константную область альфа (Cα) или ее часть и/или бета-цепь, содержащую вариабельную область бета (Vβ) и константную область бета (Cβ) или ее часть.
[0215] В некоторых случаях области Cα и Cβ являются константными областями мыши. В некоторых вариантах осуществления область Cα содержит аминокислотную последовательность, представленную в SEQ ID NO: 262 или 317, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности, такую как последовательность, имеющая по меньшей мере или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью. В некоторых случаях область Cβ содержит аминокислотную последовательность, представленную в SEQ ID NO: 263 или 109, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности, такую как последовательность, имеющая по меньшей мере или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью.
[0216] В некоторых вариантах осуществления области Cα и Cβ представляют собой константные области человека. В некоторых таких вариантах осуществления область Cα содержит аминокислотную последовательность, представленную в любой из SEQ ID NO: 212, 213, 215, 217, 218, 220 или 524, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% последовательности идентичность, такая как последовательность, имеющая по меньшей мере или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичность с такой последовательностью. В некоторых аспектах область Cβ содержит аминокислотную последовательность, представленную в SEQ ID NO: 214, 216, 631 или 889, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности, такую как последовательность, имеющая в по меньшей мере или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью.
[0217] В некоторых вариантах осуществления области Cα и/или Cβ модифицируют, например, путем включения одного или более ненативных остатков цистеина. В некоторых вариантах осуществления константная область является модифицированной формой константной области человека (например, модифицированной по сравнению с областью Cα, представленной в любой из SEQ ID NO: 212, 213, 215, 217, 218, 220 или 524, и/или областью Cβ, представленной в SEQ ID NO: 214, 216, 631 или 889. В некоторых вариантах осуществления модификацией является введение цистеина по остатку Thr48 цепи Cα и/или Ser57 цепи Cβ, по остатку Thr45 цепи Cα и/или Ser77 цепи Cβ, по остатку Tyr10 цепи Cα и/или Ser17 цепи Cβ, по остатку Thr45 цепи Cα и Asp59 цепи Cβ и/или по остатку Ser15 цепи Cα и Glu15 цепи Cβ со ссылкой на нумерацию Cα, прдставленную в любой из SEQ ID NO: 212, 213, 217, 218 или 524, или Cβ, прдставленную в SEQ ID NO: 214 или 216. Соответствующие остатки могут быть идентифицированы путем выравнивания эталонной последовательности с любой из SEQ ID NO: 212, 213, 217, 218 524 214 или 216. Например, Thr48 в цепи Cα выравнивается или соответствует Thr49 в последовательности, представленной в SEQ ID NO: 215 или 220, и Ser57 в цепи Cβ выравнивается или соответствует Ser58 в последовательности, представленной в SEQ ID NO: 631. или 889. В некоторых таких вариантах осуществления область Cα содержит ненативный цистеин в остатке 48 (или в соответствующем остатке, например в остатке 49) и содержит аминокислотную последовательность, представленную в любой из SEQ ID NO: 196, 198, 200, 201, 203, 525, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности, такую как последовательность, имеющая по меньшей мере или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью и которая содержит введенный ненативный цистеиновый остаток или остатки. В некоторых аспектах область Cβ содержит ненативный цистеин в остатке 57 (или в соответствующем остатке, например в остатке 58) и содержит аминокислотную последовательность, представленную в SEQ ID NO: 197, 199, 632, 890 или 1363, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности, такую как последовательность, имеющая, по меньшей мере, или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью и которая содержит ненативный цистеиновый остаток или остатки.
[0218] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержит альфа-цепь, содержащую последовательность аминокислот, представленную в SEQ ID NO: 18, 28, 38, 48, 58, 68, 78, 88, 98, 287 или 291, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности, такую как последовательность, имеющая, по меньшей мере, или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью, и/или бета-цепь, содержащую последовательность аминокислот, представленную в SEQ ID NO: 22, 32, 42, 52, 62, 72, 82, 92, 102, 285, 289, 293, 479, 494, 512, 526, 541, 556, 574, 589, 601, 613, 625, 639, 651, 663, 681, 696, 714, 731, 746, 764, 777, 789, 804, 820, 835, 847, 859, 871, 883, 897, 913, 927, 941, 953, 965, 977, 989, 1004 или 1376, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности, такую как последовательность, имеющая, по меньшей мере, или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью.
[0219] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержит альфа-цепь, содержащую последовательность аминокислот, представленную в SEQ ID NO: 19, 29, 39, 49, 59, 69, 79, 89, 99, 284, 288 292, 474, 489, 501, 507, 519, 533, 551, 566, 584, 596, 608, 620, 634, 646, 658, 673, 688, 706, 723, 738, 756, 772, 784, 796, 812, 827, 842, 854, 866, 878, 892, 905, 922, 934, 948, 960, 972, 984, 996 или 1387, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности, такую как последовательность, имеющая, по меньшей мере, или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью, и/или бета-цепь, содержащую последовательность аминокислот, представленную в SEQ ID NO: 23, 33, 43, 53, 63, 73, 83, 93, 103, 286, 290, 294, 480, 495, 513, 527, 542, 557, 575, 590, 602, 614, 626 640, 652, 664, 682, 697, 715, 732, 747, 765, 778, 790, 805, 821, 836, 848, 860, 872, 884, 898, 914, 928, 942, 954, 966, 978, 990, 1005 или 1377, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности, такую как последовательность, имеющая по меньшей мере или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью.
[0220] В некоторых вариантах осуществления альфа-цепь и/или бета-цепь TCR кодируется последовательностью нуклеотидов, содержащей сигнальный пептид (также называемой лидерной последовательностью). Неограничивающими примерами такого сигнального пептида являются сигнальные пептиды, которые имеют или содержат последовательность аминокислот, представленную в любой из SEQ ID NO: 181-182, 184-194, 310, 311, 487, 540, 549, 564, 573, 582, 671, 680, 695, 704, 713, 730, 745, 754, 763, 770, 803, 810, 819, 834, 903, 912, 920, 1003 или 1011. В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент кодируется последовательностью нуклеотидов, которая кодирует: а) альфа-цепь, содержащую последовательность аминокислот, представленную в SEQ ID NO: 318, 319, 322, 323, 326, 327, 330, 331, 334, 335, 338, 339, 130, 131, 134, 135, 195, 205, 222, 242, 253, 256, 313,314, 475, 476, 490, 491, 502, 503, 508, 509, 520, 521, 534, 535, 552, 553, 567, 568, 585, 586, 597, 598, 609, 610, 621, 622, 635, 636, 647, 648, 659, 660, 674, 675, 689, 690, 707, 708, 724, 725, 739, 740, 757, 758, 773, 774, 785, 786, 797, 798, 813, 814, 828, 829, 843, 844, 855, 856, 867, 868, 879, 880, 893, 894, 906, 907, 923,924, 935, 936, 949, 950, 961, 962, 973, 974, 985, 986, 997, 998, 1388, 1389, или последовательность аминокислот, которая имеет с ними по меньшей мере, 90% идентичности последовательности, такую как последовательность, имеющую, по меньшей мере, или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью, и/или b) бета-цепь, содержащую последовательность аминокислот, представленную в SEQ ID NO: 320, 321, 324, 325, 328, 329, 332, 333, 336, 337, 110, 129, 132, 133, 179, 180, 206, 221, 246, 250, 260, 312, 315,316,481, 482, 496, 497, 514, 515, 616, 528, 529, 543, 544, 558, 559, 576, 577, 591, 592, 603, 604, 615, 627, 628, 641, 642, 653, 654, 665, 666, 683, 684, 698, 699, 716, 717, 733, 734, 748, 749, 766, 767, 779, 780, 791, 792, 806, 807, 822, 823, 837, 838, 849, 850, 861, 862, 873, 874, 885, 886, 899, 900, 915, 916, 929, 930, 943, 944, 955, 956, 967, 968, 979, 980, 991, 992, 1006, 1007 или 1378 -1379, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности, такую как последовательность, имеющая, по меньшей мере, или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью. В некоторых вариантах осуществления альфа-цепь и бета-цепь могут быть связаны через линкер, такой как любой, описанный в настоящем документе в другом месте.
[0221] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент распознает или связывается с эпитопом или областью E6 HPV16, таким как пептидный эпитоп, содержащий аминокислотную последовательность, представленную в любой из SEQ ID NO: 232-234. В некоторых случаях TCR или его антигенсвязывающий фрагмент не распознает или не связывается с эпитопом E6 (29-38), содержащим аминокислотную последовательность TIHDIILECV (SEQ ID NO: 233). В некоторых случаях TCR или его антигенсвязывающий фрагмент, который распознает или связывается с пептидным эпитопом, полученным из E6 HPV16, представляет собой или содержит последовательность, представленную в SEQ ID NO: 232 или SEQ ID NO: 234.
[0222] В некоторых аспектах TCR или антигенсвязывающий фрагмент распознают или связываются с эпитопом или областью белка Е7 HPV16, таким как пептидный эпитоп, содержащий аминокислотную последовательность, представленную в любой из SEQ ID NO: 235-239. В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент не распознает или не связывается с эпитопом E7 (11-19), содержащим аминокислотную последовательность YMLDLQPET (SEQ ID NO: 236). В некоторых случаях пептид, полученный из E7 HPV16, представляет собой или содержит последовательность, представленную в SEQ ID NO: 235.
a. E6 HPV 16(29-38)
[0223] В некоторых случаях TCR распознает или связывается с пептидным эпитопом, полученным из E6 HPV16, который представляет собой или содержит E6 (29-38) TIHDIILECV (SEQ ID NO: 233). В некоторых вариантах осуществления TCR распознает или связывается с E6 HPV 16 (29-38) в контексте MHC, такого как MHC класса I, например HLA-A2. В некоторых вариантах осуществления E6 HPV 16 содержит последовательность, представленную в SEQ ID NO: 264.
[0224] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержит область Vα, которая содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность X1X2X3X4X5X6X7X8X9X10X11X12X13X14X15X16X17X18 (SEQ ID NO: 1370), где X1 представляет собой A, I или В; Х2 представляет собой М, L, S или V; Х3 представляет собой R, L, Q или N; X4 представляет собой E, V, T, P, G или F; X5 представляет собой G, I, L, A, отсутствует или P; X6 представляет собой R, T, G, отсутствует или представляет собой S; X7 представляет собой G, R или отсутствует; X8 представляет собой T, G или отсутствует; Х9 отсутствует или представляет собой А; Х10 отсутствует или представляет собой G; X11 отсутствует или представляет собой G; X12 отсутствует или представляет собой T; X13 отсутствует или представляет собой S; X14 представляет собой G, Y, отсутствует или представляет собой N; Х15 представляет собой F, G, N или Т; X16 представляет собой K, N или P; Х17 представляет собой Т или L; и X18 представляет собой I, F, V или T.
[0225] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержит область Vα, которая содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность X1X2X3X4X5X6X7X8X9X10X11X12X13X14X15X16X17X18 (SEQ ID NO: 248), где X1 представляет собой A, I или В; Х2 представляет собой М, L, S или V; Х3 представляет собой R, L, Q или N; X4 представляет собой E, V, T, P, G или F; X5 представляет собой G, I, L, A, отсутствует или P; X6 представляет собой R, T, G, отсутствует или представляет собой S; X7 представляет собой G, R или отсутствует; X8 представляет собой T, G или отсутствует; Х9 отсутствует или представляет собой А; Х10 отсутствует или представляет собой G; X11 отсутствует или представляет собой G; X12 отсутствует или представляет собой T; X13 отсутствует или представляет собой S; X14 представляет собой G, Y, отсутствует или представляет собой N; Х15 представляет собой F, G, N или Т; X16 представляет собой K, N или P; Х17 представляет собой Т или L; и X18 представляет собой I, V, F или T.
[0226] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержит область Vα, которая содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность X1X2X3X4X5X6X7X8X9X10X11X12X13X14X15X16X17X18 (SEQ ID NO: 1205), где X1 представляет собой A, I или В; X2 представляет собой M, L, A, V, S или E; X3 представляет собой R, L, N, S, Q, K, G или W; X4 представляет собой E, V, P, T, F, A, G, N, D или L; X5 представляет собой G, I, D, L, A, P, H, N, R, T или отсутствует; X6 представляет собой G, N, R, T, M, S, P или отсутствует; X7 представляет собой G, V, D, L, Q, T, R, N или отсутствует; X8 представляет собой T, D, S, L, G или отсутствует; X9 представляет собой A, G, Q или отсутствует; X10 представляет собой G или отсутствует; X11 представляет собой G или отсутствует; Х12 представляет собой Т или отсутствует; X13 представляет собой S, A, T, G или отсутствует; X14 представляет собой G, Y, T, N, A, W или отсутствует; X15 представляет собой F, G, N, T, Y, D, S, R, Q или E; X16 представляет собой K, P, A, N, D или Q; X17 представляет собой L, M, I, V или T; и X18 представляет собой I, T, V, N, F, R или Q.
[0227] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержит область Vα, которая содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность X1X2X3X4X5X6X7X8X9X10X11X12X13X14X15X16X17X18 (SEQ ID NO: 1220), где X1 представляет собой A, I или В; X2 представляет собой M, L, A, V, S или E; X3 представляет собой R, L, N, S, Q, K, G или W; X4 представляет собой E, V, P, T, F, A, G, N, D или L; X5 представляет собой G, I, D, L, A, P, N, R, T или отсутствует; X6 представляет собой G, N, R, T, M, S, P или отсутствует; X7 представляет собой G, V, D, L, Q, T, R или отсутствует; X8 представляет собой T, D, S, L, G или отсутствует; X9 представляет собой A, G, Q или отсутствует; X10 представляет собой G или отсутствует; X11 представляет собой G или отсутствует; Х12 представляет собой Т или отсутствует; X13 представляет собой S, A, T, G или отсутствует; X14 представляет собой G, Y, T, N, A, W или отсутствует; X15 представляет собой F, G, N, T, Y, D, S, R, Q или E; X16 представляет собой K, P, A, D или Q; X17 представляет собой L, M, I, V или T; и X18 представляет собой I, T, V, F, R или Q.
[0228] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержит область Vα, которая содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность X1X2X3X4X5X6X7X8X9X10X11X12X13X14X15X16LT (SEQ ID NO: 1206), где X1 представляет собой A, I или В; X2 представляет собой L, M, V или E; Х3 представляет собой L, R, N, G или S; X4 представляет собой V, T, F, N, E, P, G или L; X5 представляет собой I, A, P, N, G или T; X6 представляет собой R, G, S или T; X7 представляет собой G, R, L, V или T; X8 представляет собой T, G, L или отсутствует; X9 представляет собой A, G, Q или отсутствует; X10 представляет собой G или отсутствует; X11 представляет собой G или отсутствует; Х12 представляет собой Т или отсутствует; X13 представляет собой S, T или G; X14 представляет собой Y, A, G или N; X15 представляет собой G, S, N, R или E; и X16 представляет собой K или Q.
[0229] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержит область Vα, которая содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность AMRX4X5X6X7X8X9X10X11X12X13X14X15 (SEQ ID NO: 1207), где X4 представляет собой E, T, A, D или L; Х5 представляет собой G, A, N или R; Х6 представляет собой R, G, R, Т, М или S; X7 представляет собой G, V, D, L или отсутствует; X8 представляет собой T, D или отсутствует; X9 представляет собой G или отсутствует; X10 представляет собой S, T, G или отсутствует; X11 представляет собой G, Y, N, A или W; Х12 представляет собой F, G, N, D, S или Y; X13 представляет собой K, D, Q; Х14 представляет собой Т, L, М или I; и X15 представляет собой I, T, R или Q.
[0230] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержит область Vα, которая содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность X1X2X3X4X5X6X7X8X9X10X11X12X13X14X15KX17X18 (SEQ ID NO: 1208), где X1 представляет собой I или V; Х2 представляет собой L или V; Х3 представляет собой L, N или R; Х4 представляет собой V, F или G; X5 представляет собой I, P, G или T; X6 представляет собой R, S, P или G; X7 представляет собой G, R, Q, T или V; Х8 представляет собой Т, G, S или L; X9 представляет собой A, G, Q или отсутствует; X10 представляет собой G или отсутствует; X11 представляет собой G или отсутствует; Х12 представляет собой Т или отсутствует; X13 представляет собой G или S; Х14 представляет собой Y или N; X15 представляет собой G, Q или E; Х17 представляет собой V или L; и X18 представляет собой I или Т.
[0231] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержит область Vα, которая содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность AX2RX4AX6NNDMR (SEQ ID NO: 1221), где X2 представляет собой V или M; Х4 представляет собой Р или D; Х6 представляет собой N или R.
[0232] В некоторых вариантах осуществления область Vα содержит область 1, определяющую комплементарность, (CDR-1), содержащую аминокислотную последовательность X1X2X3X4X5X6X7 (SEQ ID NO: 1371), где X1 представляет собой T, D или N; Х2 представляет собой I или S; X3 представляет собой S, D или A; X4 представляет собой G, Q, P или отсутствует; Х5 представляет собой Т, S или I; Х6 представляет собой D, Y или Q; и X7 представляет собой Y, G, N или Q. В некоторых вариантах осуществления область Vα содержит область 1, определяющую комплементарность, (CDR-1), содержащую аминокислотную последовательность X1X2X3X4X5X6X7 (SEQ ID NO: 240), где X1 представляет собой T, D S или N; Х2 представляет собой I или S; X3 представляет собой S, D, N, Y или A; X4 представляет собой G, Q, P или отсутствует; Х5 представляет собой Т, S, F или I; Х6 представляет собой D, Y, P или Q; и X7 представляет собой Y, G, N, A, S или Q. В некоторых вариантах осуществления область Vα содержит область 1, определяющую комплементарность, (CDR-1), содержащую аминокислотную последовательность X1X2X3X4X5X6X7 (SEQ ID NO: 1209), где X1 представляет собой T, N, D или S; Х2 представляет собой S, I или R; X3 представляет собой D, S, M, A, Y, N или G; X4 представляет собой Q, G, P или отсутствует; X5 представляет собой S, T, F, I или N; X6 представляет собой Y, D, Q, P, N или E; и X7 представляет собой G, Y, N, S или A.
[0233] В некоторых примерах область Vα содержит область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность X1X2X3X4X5X6X7X8 (SEQ ID NO: 1372), где X1 представляет собой G, Q, I или V; Х2 представляет собой L, S, Q или Y; Х3 представляет собой Т, G или S; X4 представляет собой Y, S или отсутствует; Х5 отсутствует или представляет собой D; X6 отсутствует, E, Q или S; X7 представляет собой S, Q, R или G; и X8 представляет собой N или E. В некоторых примерах область Vα содержит область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность X1X2X3X4X5X6X7X8 (SEQ ID NO: 244), где X1 представляет собой G, Q, I, M, Y или V; Х2 представляет собой L, S, Q, T или Y; Х3 представляет собой Т, G, L или S; X4 представляет собой Y, S, N, A или отсутствует; X5 отсутствует, A или D; X6 отсутствует, E, Q, T или S; X7 представляет собой S, Q, R, L или G; и X8 представляет собой N, V или E. В некоторых примерах область Vα содержит область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность X1X2X3X4X5X6X7X8 (SEQ ID NO: 1210), где X1 представляет собой Q, G, I, V, Y, M, R или N; X2 представляет собой G, L, S, Q, Y, T, N или V; X3 представляет собой S, T, L или K; X4 представляет собой Y, I, S, A, N, F или отсутствует; X5 представляет собой D, A или отсутствует; X6 представляет собой E, K, Q, S, T, G, D или отсутствует; X7 представляет собой Q, S, N, R, G, L или D; и X8 представляет собой N, K, E, V или L.
[0234] В некоторых аспектах TCR или его антигенсвязывающий фрагмент содержит область Vβ, которая содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность ASSX4X5X6X7X8X9X10X11X12X13 (SEQ ID NO: 1373), где X4 представляет собой H, P, L или Y; Х5 представляет собой L, G, W, F или S; X6 представляет собой A, G или L; X7 представляет собой G, E, A, T или отсутствует; X8 представляет собой F, G, T или S; Х9 представляет собой Т, N, Н или А; X10 представляет собой G, T, Q, D или Y; X11 представляет собой E, P, T или G; X12 представляет собой L, A, Q или Y; и X13 представляет собой F, H, Y или T.
[0235] В некоторых аспектах TCR или его антигенсвязывающий фрагмент содержит область Vβ, которая содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность ASX3X4X5X6X7X8X9X10X11X12X13 (SEQ ID NO: 258), где X3 представляет собой S или T; Х4 представляет собой Н, Р, L, F или Y; Х5 представляет собой L, G, W, F, T или S; X6 представляет собой A, G или L; X7 представляет собой G, E, A, T, Q или отсутствует; X8 представляет собой F, G, T, R или S; X9 представляет собой T, N, H, R, E или A; X10 представляет собой G, T, Q, D, R или Y; X11 представляет собой E, P, T или G; X12 представляет собой L, A, Q или Y; и X13 представляет собой F, H, Y или T.
[0236] В некоторых аспектах TCR или его антигенсвязывающий фрагмент содержит область Vβ, которая содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность X1X2X3X4X5X6X7X8X9X10X11X12X13X14X15 (SEQ ID NO: 1211), где X1 представляет собой A, S или В; X2 представляет собой S, A или V; X3 представляет собой S, V, R или Q; X4 представляет собой H, P, Q, L, Y, G, T, F, S, R или E; X5 представляет собой L, G, R, W, F, S, V, T, Y, Q или отсутствует; X6 представляет собой A, G, L, T, E, P или отсутствует; X7 представляет собой G, T, A, R, Q, N, S или отсутствует; X8 представляет собой G, S или отсутствует; X9 представляет собой G или отсутствует; X10 представляет собой F, G, A, S, T, R, Q, L или отсутствует; X11 представляет собой T, N, F, A, R, S, G или отсутствует; X12 представляет собой G, T, L D, Y, N, Q, S или E; X13 представляет собой E, W, T, G, K, N или P; X14 представляет собой L, A, K, Q, Y или I; и X15 представляет собой F, H, Y, T или I.
[0237] В некоторых аспектах TCR или его антигенсвязывающий фрагмент содержит область Vβ, которая содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность X1X2X3X4X5X6X7X8X9X10X11X12X13X14X15 (SEQ ID NO: 1222), где X1 представляет собой A, S или В; X2 представляет собой S, A или V; X3 представляет собой S, R или Q; X4 представляет собой H, P, Q, L, Y, G, T, F, S, R или E; X5 представляет собой L, G, R, W, F, S, V, T, Y, Q или отсутствует; X6 представляет собой A, G, L, E, P или отсутствует; X7 представляет собой G, T, A, R, Q, N, S или отсутствует; X8 представляет собой G, S или отсутствует; X9 представляет собой G или отсутствует; X10 представляет собой F, G, A, S, T, R, Q, L или отсутствует; X11 представляет собой T, N, F, A, R, S, G или отсутствует; X12 представляет собой G, T, L D, Y, N, Q, S или E; X13 представляет собой E, W, T, G, K, N или P; X14 представляет собой L, A, K, Q, Y или I; и X15 представляет собой F, H, Y, T или I.
[0238] В некоторых аспектах TCR или его антигенсвязывающий фрагмент содержит область Vβ, которая содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность ASSX4X5X6X7X8X9X10X11X12X13X14 (SEQ ID NO: 1212), где X4 представляет собой H, P, Q, L, Y, F, R или E; X5 представляет собой L, G, R, W, F, S, V, T, Y или Q; X6 представляет собой A, G, L, E P; X7 представляет собой G, T, A, R, Q, S или отсутствует; X8 представляет собой G, S или отсутствует; X9 представляет собой F, G, A, S, T, R, L или отсутствует; Х10 представляет собой Т, N, А, F, R, S или G; X11 представляет собой G, T, L, D, Y, Q, S, E или N; X12 представляет собой E, W, T, G, P, K; X13 представляет собой L, A, K, Q, Y или I; и X14 представляет собой F, H, Y или T.
[0239] В некоторых аспектах TCR или его антигенсвязывающий фрагмент содержит область Vβ, которая содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность X1X2X3X4X5X6X7X8X9X10X11X12X13QY (SEQ ID NO: 1213), где X1 представляет собой A или S; X2 представляет собой S, V или A; Х3 представляет собой S или V; Х4 представляет собой L, Y, P или S; Х5 представляет собой W, F, V, L или Y; X6 представляет собой G, T или A; X7 представляет собой A, R, Q, S или отсутствует; X8 представляет собой G или отсутствует; X9 представляет собой G или отсутствует; X10 представляет собой S, T, R или G; X11 представляет собой T, A, R, S или N; X12 представляет собой D, Y, T или G; и X13 представляет собой T или E.
[0240] В некоторых аспектах TCR или его антигенсвязывающий фрагмент содержит область Vβ, которая содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность X1X2SX4X5X6X7X8X9X10X11X12X13QY (SEQ ID NO: 1223), где X1 представляет собой A или S; Х2 представляет собой S или А; Х4 представляет собой L, Y, P или S; Х5 представляет собой W, F, V, L или Y; X6 представляет собой G или A; X7 представляет собой A, R, Q, S или отсутствует; X8 представляет собой G или отсутствует; X9 представляет собой G или отсутствует; X10 представляет собой S, T, R или G; X11 представляет собой T, A, R, S или N; X12 представляет собой D, Y, T или G; и X13 представляет собой T или E.
[0241] В некоторых аспектах TCR или его антигенсвязывающий фрагмент содержит область Vβ, которая содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность ASX3X4X5X6X7X8X9X10X11X12F (SEQ ID NO: 1214), где X3 представляет собой S, Q или Р; Х4 представляет собой Н, Р, Т или Е; Х5 представляет собой L, G, W или F; X6 представляет собой A, G или отсутствует; X7 представляет собой G, N, S, R или отсутствует; X8 представляет собой F, G, Q, L, A или отсутствует; Х9 представляет собой Т, N или А; X10 представляет собой G, T, N или E; X11 представляет собой E, N или K; и X12 представляет собой L, A или Q.
[0242] В некоторых аспектах TCR или его антигенсвязывающий фрагмент содержит область Vβ, которая содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность ASSX4X5X6X7X8NYX11YT (SEQ ID NO: 1215), где X4 представляет собой L или R; Х5 представляет собой S или Т; X6 представляет собой G, T или A; Х7 представляет собой Т или отсутствует; X8 представляет собой G или отсутствует; и X11 представляет собой G или отсутствует.
[0243] В некоторых аспектах TCR или его антигенсвязывающий фрагмент содержит область Vβ, которая содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность ASSX4WGX7SNQPX12H (SEQ ID NO: 1216), где X4 представляет собой L, F или P; Х7 представляет собой R или Q; и X12 представляет собой Q или L.
[0244] В некоторых аспектах TCR или его антигенсвязывающий фрагмент содержит область Vβ, которая содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность ASSX4X5X6X7X8SGNTIY (SEQ ID NO: 1217), где X4 представляет собой L или R; Х5 представляет собой W или Q; X6 представляет собой G или P; Х7 представляет собой R или S; и X8 представляет собой S или отсутствует.
[0245] В некоторых случаях область Vβ содержит область 1, определяющую комплементарность, (CDR-1), содержащую аминокислотную последовательность X1X2HX4X5 (SEQ ID NO: 252), где X1 представляет собой S или M; X2 представляет собой G, E, D или N; X4 представляет собой V, N или E; и X5 представляет собой S, R, N или Y. В некоторых случаях область Vβ содержит область 1, определяющую комплементарность, (CDR-1), содержащую аминокислотную последовательность X1X2X3X4X5X6 (SEQ ID NO: 1218), где X1 представляет собой S, M, D или L; X2 представляет собой G, E, D, N, Q, S или F; X3 представляет собой H, V, Y, N или Q; X4 представляет собой A, S, F или отсутствует; X5 представляет собой W V, N, E, T, P, Y, K, D или L; и X6 представляет собой S, R, A, N, Y, M или T.
[0246] В некоторых случаях область Vβ содержит область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность X1X2X3X4X5X6 (SEQ ID NO: 255), где X1 представляет собой F или S; Х2 представляет собой Q, Y или V; Х3 представляет собой N, D или G; Х4 представляет собой Е или V; Х5 представляет собой А, К или G; и X6 представляет собой Q, M или T. В некоторых случаях область Vβ содержит область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность X1X2X3X4X5X6X7 (SEQ ID NO: 1219), где X1 представляет собой F, Y, S AM; X2 представляет собой N, Q, V, T, Y или A; X3 представляет собой N, D, E, S, G, I, F, Q или L; X4 представляет собой G, A, N или отсутствует; Х5 представляет собой Е, К, V, Е, S, Т, G или N; X6 представляет собой A, E, K, G, L, D, V или N; и X7 представляет собой Q, M, T, A, V, E, P, D или I.
[0247] В некоторых вариантах осуществления область Vα содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 138, 144, 147, 163, 167, 173, 304, 308, 478, 493, 505, 511, 523, 539, 555, 572, 588, 600, 612, 624, 638, 650, 662 или 679, или последовательность, имеющую по меньшей мере или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью. В некоторых примерах область Vα содержит CDR3, содержащуюся в аминокислотной последовательности, представленной в любой из SEQ ID NO: 111, 113, 115, 121, 123, 125, 297, 299, 477, 492, 504, 510, 522, 536, 554, 569, 587, 599, 611, 623, 637, 649, 661 или 676, или последовательности, имеющей по меньшей мере или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98, или 99% идентичности с такой последовательностью. В некоторых вариантах осуществления область Vα дополнительно содержит область 1, определяющую комплементарность, (CDR-1), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 136, 142, 161, 165 171, 302, 306, 537, 570, или 677, или последовательность, имеющую по меньшей мере или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью. В некоторых аспектах область Vα содержит CDR-1, содержащуюся в аминокислотной последовательности, представленной в любой из SEQ ID NO: 111, 113, 115, 121, 123, 125, 297, 299, 477, 492, 504, 510, 522, 536, 554, 569, 587, 599, 611, 623, 637, 649, 661 или 676, или последовательности, имеющей по меньшей мере или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью. В некоторых вариантах осуществления область Vα дополнительно содержит область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 137, 143, 162, 166, 172, 303, 307, 538, 571 или 678, или последовательность, имеющую, по меньшей мере, или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью. В некоторых случаях область Vα содержит CDR-2, содержащуюся в аминокислотной последовательности, представленной в любой из SEQ ID NO: 111, 113, 115, 121, 123, 125, 297, 299, 477, 492, 504, 510, 522, 536, 554, 569, 587, 599, 611, 623, 637, 649, 661 или 676, или последовательности, имеющей по меньшей мере или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью.
[0248] В некоторых вариантах осуществления область Vβ содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 141, 146, 150, 164, 170 174, 305, 309, 486, 499, 517, 531, 548, 563, 581, 594, 606, 618, 630, 644, 656, 670 или 686, или CDR3, содержащуюся в аминокислотной последовательности, представленной в любой из SEQ ID NO: 112, 114, 116, 122, 124 126, 298, 300, 483, 498, 516, 530, 545, 560, 578, 593, 605, 617, 629, 643, 655, 667 или 685, или последовательности, имеющей по меньшей мере или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью. В некоторых вариантах осуществления область Vβ содержит область 1, определяющую комплементарность, (CDR-1), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 139, 145, 148, 168, 484, 546, 561, 579 или 668, или последовательность, имеющуюей по меньшей мере или примерно 90, 91, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью. В некоторых случаях область Vβ содержит CDR-1, содержащуюся в аминокислотной последовательности, представленной в любой из SEQ ID NO: 112, 114, 116, 122, 124 126, 298, 300, 483, 498, 516, 530, 545, 560, 578, 593, 605, 617, 629, 643, 655, 667 или 685, или последовательности, имеющей по меньшей мере или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98, или 99% идентичности с такой последовательностью. В некоторых вариантах осуществления область Vβ дополнительно содержит область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 140, 149, 169, 485, 547, 562, 580 или 669, или последовательность, имеющую, по меньшей мере, 90 или 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью. В некоторых примерах область Vβ содержит CDR-2, содержащуюся в аминокислотной последовательности, представленной в любой из SEQ ID NO: 112, 114, 116, 122, 124, 126, 298, 300, 483, 498, 516, 530, 545, 560, 578, 593, 605, 617, 629, 643, 655, 667 или 685, или последовательности, имеющей по меньшей мере или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98, или 99% идентичности с такой последовательностью.
[0249] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержит область Vα, которая содержит область 1, определяющую комплементарность, (CDR-1), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 136, 142, 161, 165, 171, 302, 306,537, 570 или 677, область 2, определяющую комплементарность, (CDR-2), содержащая аминокислотную последовательность, представленную в любой из SEQ ID NO: 137, 143, 162, 166, 172, 303, 307,538, 571 или 678, и/или область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 138, 144, 147, 163, 167 173, 304, 308, 478, 493, 505, 511, 523, 539, 555, 572, 588, 600, 612, 624, 638, 650, 662 или 679. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99%, идентичные таким последовательностям. В некоторых аспектах TCR или антигенсвязывающий его фрагмент содержит область Vβ, которая содержит область 1, определяющую комплементарность, (CDR-1), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 139, 145, 148, 168, 484, 546, 561, 579 или 668, область, определяющую комплементарность 2 (CDR-2), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 140, 149, 169, 485, 547, 562, 580 или 669, и/или область, определяющую комплементарность 3 (CDR-3), содержащий аминокислотную последовательность, представленную в любой из SEQ ID NO: 141, 146, 150, 164, 170 174, 305, 309, 486, 499, 517, 531, 548, 563, 581, 594, 606, 618, 630, 644, 656, 670 или 686. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0250] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержит область Vα, которая содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 136, 137 и 138, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 139, 140 и 141, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0251] В некоторых вариантах осуществления область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 142, 143 и 144, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 145, 140 и 146, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0252] В некоторых вариантах осуществления область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 136, 137 и 147, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 150, соответственно. Также среди представленных TCR имеются такие, которые содержат последовательности, по меньшей мере, или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0253] В некоторых вариантах осуществления область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 161, 162 и 163, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 164, соответственно. Также среди представленных TCR имеются такие, которые содержат последовательности, по меньшей мере, или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0254] В некоторых вариантах осуществления область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 165, 166 и 167, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 168, 169 и 170, соответственно. Также среди представленных TCR имеются такие, которые содержат последовательности, по меньшей мере, или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0255] В некоторых вариантах осуществления область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 171, 172 и 173, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 174, соответственно. Также среди представленных TCR имеются такие, которые содержат последовательности, по меньшей мере, или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0256] В некоторых вариантах осуществления область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 302, 303 и 304, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 139, 140 и 305, соответственно. Также среди представленных TCR имеются такие, которые содержат последовательности, по меньшей мере, или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0257] В некоторых вариантах осуществления область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 306, 307 и 308, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 309, соответственно. Также среди представленных TCR имеются такие, которые содержат последовательности, по меньшей мере, или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0258] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержит область Vα, которая содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 136, 137 и 478, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 484, 485 и 486, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0259] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержит область Vα, которая содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 161, 162 и 493, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 499, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0260] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержит область Vα, которая содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 165, 166 и 505, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 499, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0261] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержит область Vα, которая содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 161, 162 и 511, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 517, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0262] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержит область Vα, которая содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 136, 137 и 523, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 531, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0263] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержит область Vα, которая содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 537, 538 и 539, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 546, 547 и 548, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0264] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержит область Vα, которая содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 136, 137 и 555, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 561, 562 и 563, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0265] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержит область Vα, которая содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 570, 571 и 572, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 579, 580 и 581, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0266] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержит область Vα, которая содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 136, 137 и 588, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 594, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0267] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержит область Vα, которая содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 136, 137 и 600, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 606, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0268] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержит область Vα, которая содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 136, 137 и 612, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 618, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0269] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержит область Vα, которая содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 136, 137 и 624, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 168, 169 и 630, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0270] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержит область Vα, которая содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 142, 143 и 638, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 561, 562 и 644, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0271] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержит область Vα, которая содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 171, 172 и 650, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 656, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0272] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержит область Vα, которая содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 136, 137 и 662, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 668, 669 и 670, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0273] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержит область Vα, которая содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 677, 678 и 679, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 686, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0274] В некоторых вариантах осуществления область Vα содержит область 1, определяющую комплементарность, (CDR-1), CDR-2 и CDR-3, соответственно, содержащие аминокислотные последовательности CDR-1, CDR-2 и CDR-3, содержащиеся в аминокислотной последовательности области Vα, представленной в любой из SEQ ID NO: 111, 113, 115, 121, 123, 125, 297, 299, 477, 492, 504, 510, 522, 536, 554, 569, 587, 599, 611, 623, 637, 649, 661 или 676. В некоторых аспектах область Vβ содержит область 1, определяющую комплементарность, (CDR-1), CDR-2 и CDR-3, соответственно, содержащие аминокислотные последовательности CDR-1, CDR-2 и CDR-3, содержащиеся в аминокислотной последовательности области Vβ, представленной в любой из SEQ ID NO: 112, 114, 116, 122, 124, 126, 298, 300, 483, 498, 516, 530, 545, 560, 578, 593, 605, 617, 629, 643, 655, 667 или 685. Также среди представленных TCR имеются такие, которые содержат последовательности, по меньшей мере, или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0275] В некоторых вариантах осуществления TCR или антигенсвязывающий фрагмент включают область Vα, которая содержит область 1, определяющую комплементарность, (CDR-1), CDR-2 и CDR-3, соответственно содержащие аминокислотные последовательности CDR-1, CDR-2, и CDR-3, представленные в Таблице 2; и область Vβ, которая содержит область 1, определяющую комплементарность, (CDR-1), CDR-2 и CDR-3, соответственно, содержащие аминокислотные последовательности CDR-1, CDR-2 и CDR-3, представленные в Таблице 2. Также среди представленных TCR имеются такие, которые содержат последовательности, по меньшей мере, или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям. Иллюстративные TCR, содержащие такие CDR, или их модифицированные версии, как описано в другом месте настоящего документа, также представлены в Таблице 2.
TCR
[0276] В некоторых случаях TCR или его антигенсвязывающий фрагмент содержит области Vα и Vβ, содержащие аминокислотные последовательности SEQ ID NO: 111 и 112, соответственно. В некоторых вариантах осуществления области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 113 и 114, соответственно. В некоторых случаях области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 115 и 116, соответственно. В некоторых вариантах осуществления области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 121 и 122, соответственно. В некоторых аспектах области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 123 и 124, соответственно. В некоторых примерах области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 125 и 126, соответственно. В некоторых примерах области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 297 и 298, соответственно. В некоторых примерах области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 299 и 300, соответственно. В некоторых вариантах осуществления области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 477 и 483, соответственно. В некоторых примерах области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 492 и 498, соответственно. В некоторых случаях области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 504 и 498, соответственно. В некоторых случаях TCR или его антигенсвязывающий фрагмент содержат области Vα и Vβ, содержащие аминокислотные последовательности SEQ ID NO: 510 и 516, соответственно. В некоторых вариантах осуществления области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 522 и 530, соответственно. В некоторых примерах области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 536 и 545, соответственно. В некоторых случаях области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 554 и 560, соответственно. В некоторых случаях TCR или его антигенсвязывающий фрагмент содержат области Vα и Vβ, содержащие аминокислотные последовательности SEQ ID NO: 569 и 578, соответственно. В некоторых вариантах осуществления области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 587 и 593, соответственно. В некоторых примерах области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 599 и 605, соответственно. В некоторых вариантах осуществления области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 611 и 617, соответственно. В некоторых случаях области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 623 и 629, соответственно. В некоторых случаях области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 637 и 643, соответственно. В некоторых случаях области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 649 и 655, соответственно. В некоторых примерах области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 661 и 667, соответственно. В некоторых случаях области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 676 и 685, соответственно. Также среди представленных TCR имеются такие, которые содержат последовательности, по меньшей мере, или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0277] В некоторых вариантах осуществления альфа-цепь TCR или его антигенсвязывающий фрагмент дополнительно содержат область Cα или ее часть, и/или бета-цепь дополнительно содержит область Cβ или ее часть. В некоторых вариантах осуществления область Cα или ее часть содержит аминокислотную последовательность, представленную в любой из SEQ ID NO: 212, 213, 215, 218 или 524, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности, такая как последовательность, имеющая по меньшей мере или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью. В некоторых аспектах область Cβ содержит аминокислотную последовательность, представленную в SEQ ID NO: 214, 216 или 631, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности, такая как последовательность, имеющая по меньшей мере или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью. В некоторых вариантах осуществления области Cα и/или Cβ модифицируют, например, путем включения одного или более ненативных остатков цистеина, таких как любые, описанные в настоящем документе. В некоторых вариантах осуществления область Cα или ее часть содержат ненативный цистеин в остатке 48 и содержит аминокислотную последовательность, представленную в любой из SEQ ID NO: 196, 198, 201, 203 или 525, или последовательность аминокислот, которая имеет с ними, по меньшей мере, 90% идентичности последовательности, такую как последовательность, имеющая, по меньшей мере, или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью и которая содержит введенный ненативный цистеиновый остаток (например, Cys48). В некоторых аспектах область Cβ содержит ненативный цистеин в остатке 57 и содержит аминокислотную последовательность, представленную в SEQ ID NO: 197, 199 или 632, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности, например, последовательность, имеющая, по меньшей мере или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью.
[0278] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат альфа-цепь, содержащую последовательность аминокислот, представленную в SEQ ID NO: 18, 28, 38, 68, 78, 88, 287, 291, 473, 488, 500, 506, 518, 532, 550, 565, 583, 595, 607, 619, 633, 645, 657 или 672, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности, такая как последовательность, имеющая по меньшей мере или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью, и/или бета-цепь, содержащую последовательность аминокислот, представленную в SEQ ID NO: 22, 32, 42, 72, 82, 92, 289, 293, 479, 494, 512, 526, 541, 556, 574, 589, 601, 613, 625, 639, 651, 663 или 681, или последовательность аминокислот, которая имеет с ними, по меньшей мере, 90% идентичности последовательности, такая как последовательность, имеющая, по меньшей мере или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью.
[0279] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержит альфа-цепь, содержащую последовательность аминокислот, представленную в SEQ ID NO: 19, 29, 39, 69, 79, 89, 288, 292, 474, 489, 501 507, 519, 533, 551, 566, 584, 596, 608, 620, 634, 646, 658 или 673, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности, такую как последовательность, имеющая в по меньшей мере или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью, и/или бета-цепь, содержащую последовательность аминокислот, представленную в SEQ ID NO: 23, 33, 43, 73, 83, 93, 290, 294, 480, 495, 513, 527, 542, 557, 575, 590, 602, 614, 626, 640, 652, 664 или 682, или последовательность аминокислот, которая имеет с ними, по меньшей мере, 90% идентичности последовательности, такая как последовательность, имеющая, по меньшей мере или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью.
[0280] В некоторых вариантах осуществления области Vα и Vβ содержат аминокислотные последовательности, соответствующие SEQ ID NO, представленные в Таблице 3 или Таблице 4. В некоторых аспектах TCR содержит последовательности константной области альфа и константной области бета, такие как последовательности, соответствующие SEQ ID NO, представленные в Таблице 3 или Таблице 4. В некоторых случаях TCR содержит полную последовательность, содержащую вариабельную и константную области цепи, такую как последовательность, соответствующая SEQ ID NO, представленным в Таблицах 3 или 4 («Полная»). В некоторых вариантах осуществления полная последовательность, содержащая вариабельную и константную области, также включает сигнальную последовательность и, таким образом, содержит последовательность, соответствующую SEQ ID NO, представленным в Таблице 3 или 4 («Полная+сигнальная»). Также среди представленных TCR имеются такие, которые содержат последовательности, по меньшей мере, или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям. Иллюстративные TCR, содержащие такие последовательности, или их модифицированные версии, как описано в другом месте настоящего документа, также представлены в Таблицах 3 и 4, соответственно.
(Vβ)
(Vβ)
b. HPV 16 Е7 (11-19)
[0281] В некоторых случаях TCR распознает или связывает пептидный эпитоп, полученный из E7 HPV 16, который представляет собой или содержит E7 (11-19) YMLDLQPET (SEQ ID NO: 236). В некоторых вариантах осуществления TCR распознает или связывает E7 HPV 16 (11-19) в контексте MHC, такого как MHC класса I, например, HLA-A2. В некоторых вариантах осуществления E7 HPV 16 содержит последовательность, представленную в SEQ ID NO: 265.
[0282] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vα, содержащую область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность X1X2X3X4X5X6X7X8X9X10X11 (SEQ ID NO: 249), где X1 представляет собой A или V; Х2 представляет собой Е или V; X3 представляет собой S или P; Х4 представляет собой I, S или R; Х5 представляет собой R, G или D; Х6 представляет собой G, A или N; X7 представляет собой F, отсутствует или Y; Х8 представляет собой G или Т; Х9 представляет собой Т, Q или N; X10 представляет собой V, K или N; X11 представляет собой L или F и X12 представляет собой H, I или V. В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vα, содержащую область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность X1X2SX4X5X6X7X8X9X10X11 (SEQ ID NO: 1374), где X1 представляет собой A или V; Х2 представляет собой Е или V; Х4 представляет собой I или R; Х5 представляет собой R или D; Х6 представляет собой G или N; Х7 представляет собой F или Y; Х8 представляет собой N или Q; Х9 представляет собой V или N; Х10 представляет собой L или F; и X11 представляет собой H или V.
[0283] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержfт область Vα, содержащую область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность X1X2X3X4X5X6X7X8X9X10X11X12X13X14 (SEQ ID NO: 1183), где X1 представляет собой V или A; X2 представляет собой V, A, G, Q, M или E; X3 представляет собой S, G, A, N, Y, R, T или P; X4 представляет собой E, A, S, G, R.F, N, D, V, P, L, I или M; X5 представляет собой R, N, H, T, D, G, S, A, P, L, Q или F; X6 представляет собой G, H, N, A, S, L, T или отсутствует; X7 представляет собой T, S, G или отсутствует; X8 представляет собой G или отсутствует; X9 представляет собой G, Y, N, S или отсутствует; X10 представляет собой T, G, S, D, F, Y, A, N или отсутствует; X11 представляет собой Y, F, Y, Q, N или R; X12 представляет собой N, K, Q или D; X13 представляет собой Y, L, T, F, M или V; и X14 представляет собой I, T, S, V, R или Y.
[0284] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vα, содержащую область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность VVX3X4X5X6X7X8GX10X11X12X13 (SEQ ID NO: 1184), где X3 представляет собой S, N или T; Х4 представляет собой R или F; Х5 представляет собой D или А; Х6 представляет собой N или L; Х7 представляет собой Т или отсутствует; Х8 представляет собой Y или G; Х10 представляет собой Q или F; X11 представляет собой N или K; Х12 представляет собой F или Т; и Х13 представляет собой V или I.
[0285] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vα, содержащую область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность AX2X3X4X5X6X7X8X9X10X11X12X13X14 (SEQ ID NO: 1185), где X2 представляет собой A, G, V, Q, M или E; X3 представляет собой S, G, N, A, Y, R или P; X4 представляет собой E, S, A, G, F, N, D, V, P, L, I, M или R; X5 представляет собой R, N, H, T, D, G, S, P, L, Q или F; X6 представляет собой G, H, A, S, T или отсутствует; X7 представляет собой T, S, G или отсутствует; X8 представляет собой G или отсутствует; X9 представляет собой G, N, S или отсутствует; X10 представляет собой T, G, S, D, F, Y, A или N; X11 представляет собой Y, F, Q, R или N; X12 представляет собой K, Q или D; X13 представляет собой Y, L, T, M, F или V; и X14 представляет собой I, T, S, R, Y или V.
[0286] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vα, содержащую область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность X1X2X3X4X5X6X7X8X9X10KX12I (SEQ ID NO: 1186), где X1 представляет собой A или V; Х2 представляет собой А, V или Е; X3 представляет собой S, N, T, R или P; X4 представляет собой E, A, G, F, V, P, I, D или S; X5 представляет собой R, H, T, A P, S, G или F; X6 представляет собой G, H, L, T, S, A или отсутствует; X7 представляет собой S, T или отсутствует; X8 представляет собой G или отсутствует; X9 представляет собой G, T или отсутствует; Х10 представляет собой F, Y или N; и X12 представляет собой Y, T или L.
[0287] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vα, содержащую область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность AX2X3X4X5X6X7X8X9YKYI (SEQ ID NO: 1187), где X2 представляет собой A, V или E; X3 представляет собой S, N или R; X4 представляет собой E, G, V, P, I или D; X5 представляет собой R, T, P, S, G или F; X6 представляет собой G, T, S или отсутствует; X7 представляет собой S или отсутствует; X8 представляет собой G или отсутствует; и X9 представляет собой T или отсутствует.
[0288] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vα, содержащую область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность AX2X3X4X5X6X7X8X9X10X11X12X13X14 (SEQ ID NO: 1188), где X2 представляет собой G, V, Q, или М; X3 представляет собой G, A, Y, S, N или R; X4 представляет собой S, G, L, I, M или R; Х5 представляет собой N, D, G, S, L, Q или R; X6 представляет собой A, S, G или отсутствует; X7 представляет собой G или отсутствует; X8 представляет собой G или отсутствует; X9 представляет собой G, N, S или отсутствует; X10 представляет собой S, D, Y, A, N или отсутствует; X11 представляет собой Y, Q или R; Х12 представляет собой К или Q; Х13 представляет собой L или V; и X14 представляет собой S, T или V.
[0289] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vα, содержащую область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность AX2X3X4X5X6X7X8X9X10X11X12X13T (SEQ ID NO: 1189), где X2 представляет собой G, V или Q; X3 представляет собой G, Y, S или N; Х4 представляет собой S, L или М; Х5 представляет собой N, G, L или R; X6 представляет собой A, S, G или отсутствует; X7 представляет собой G или отсутствует; X8 представляет собой G или отсутствует; X9 представляет собой G, S или отсутствует; X10 представляет собой S, Y, A, N или отсутствует; X11 представляет собой Y, Q или R; Х12 представляет собой К или Q; и X13 представляет собой L или V.
[0290] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vα, содержащую область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность AX2X3X4X5X6X7YKLS (SEQ ID NO: 1190), где X2 представляет собой G или V; Х3 представляет собой А или Y; X4 представляет собой G, S или R; Х5 представляет собой D или S; X6 представляет собой N или отсутствует; и X7 представляет собой D или отсутствует.
[0291] В некоторых вариантах осуществления область Vα содержит область 1, определяющую комплементарность, (CDR-1), содержащую аминокислотную последовательность X1SX3X4X5X6 (SEQ ID NO: 241), где X1 представляет собой D или V; Х3 представляет собой S или Р; Х4 представляет собой S или F; Х5 представляет собой Т или S; и X6 представляет собой Y или N. В некоторых вариантах осуществления область Vα содержит область 1, определяющую комплементарность, (CDR-1), содержащую аминокислотную последовательность X1X2X3X4X5X6 (SEQ ID NO: 1191), где X1 представляет собой N, S, D, T, или V; X2 представляет собой S, V, R, T или I; Х3 представляет собой М, F, G, S, N, А, L, V или Р; X4 представляет собой F, S, N, A или отсутствует; X5 представляет собой D, S, Q, Y, N, V, T или P; и X6 представляет собой Y, S, R, N, G или T.
[0292] В некоторых случаях область Vα содержит область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность X1X2X3X4X5X6X7 (SEQ ID NO: 245), где X1 представляет собой I или M; Х2 представляет собой F или Т; Х3 является S или F; Х4 представляет собой N или S; Х5 представляет собой М или Е; Х6 представляет собой D или N; и X7 представляет собой M или T. В некоторых вариантах осуществления область Vα содержит область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность X1X2X3X4X5X6X7X8 (SEQ ID NO: 1192), где X1 представляет собой I, V, L, G, N, T, Y или M; X2 представляет собой S, V, Y, L, P, F, I или T; X3 представляет собой S, Y, K, L, T или F; X4 представляет собой I, G, N, A, S или отсутствует; X5 представляет собой S, D или отсутствует; X6 представляет собой K, G, N, S, D, T или E; X7 представляет собой D, E, G, A, K, L или N; и X8 представляет собой K, V, D, P, N, T, L или M.
[0293] В некоторых аспектах TCR или его антигенсвязывающий фрагмент содержат область Vβ, содержащую область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность AX2TX4RX6X7YX9X10X11 (SEQ ID NO: 259), где X2 представляет собой S или I; Х4 представляет собой Т или D; Х6 представляет собой S или Т; Х7 представляет собой S или N; Х9 представляет собой Е или G; Х10 представляет собой Q или Y; и X11 представляет собой Y или T.
[0294] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vβ, содержащую область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность AX2X3X4X5X6X7X8X9X10X11X12X13X14 (SEQ ID NO: 1193), где X2 представляет собой S, M, I, К или V; X3 представляет собой S, T, N или A; X4 представляет собой R, V P, S, T, G, L, A, I или D; X5 представляет собой F, G, R, Y, S, L, V или T; X6 представляет собой L, G, D, A, S, T, V, R или отсутствует; X7 представляет собой G, D, R, S, T или отсутствует; X8 представляет собой S или отсутствует; X9 представляет собой S, H, G, R, V, T, D, L или отсутствует; X10 представляет собой T, S, A, Y, N, G или P; X11 представляет собой D, Y, N, E, K или G; Х12 представляет собой Т, Е, G или К; X13 представляет собой Q, Y, A или L; и X14 представляет собой Y, F, T или I.
[0295] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vβ, содержащую область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность AX2TX4X5X6X7X8X9X10X11X12 (SEQ ID NO: 1194), где X2 представляет собой S, M, I, или К; X4 представляет собой P, T, G, A, S или D; Х5 представляет собой R или S; X6 представляет собой D, G, S, T или V; X7 представляет собой R, S или отсутствует; Х8 представляет собой Т, Y, G, N или S; X9 представляет собой Y, N или K; Х10 представляет собой Е или G; X11 представляет собой Q, A или Y; и X12 представляет собой Y, F или T.
[0296] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vβ, содержащую область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность AX2X3X4X5X6X7X8X9X10X11X12X13X14 (SEQ ID NO: 1195), где X2 представляет собой S, M, I, или К; X3 представляет собой S, T, A или N; X4 представляет собой R, V, S, P, T, G, L или A; X5 представляет собой F, G, R, Y, S, V или T; X6 представляет собой L, G, D, A, S, T, V или отсутствует; X7 представляет собой G, D, R, T или отсутствует; X8 представляет собой S или отсутствует; X9 представляет собой S, H, G, R, V, T, L или отсутствует; X10 представляет собой T, S, Y, A, N, G или P; X11 представляет собой D, Y, N, K, E или G; Х12 представляет собой Т или Е; X13 представляет собой Q, A или L; и X14 представляет собой Y или F.
[0297] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vβ, содержащую область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность AX2X3X4X5X6X7X8X9X10X11QY (SEQ ID NO: 1196), где X2 представляет собой S, M, I, или К; X3 представляет собой S, T, A или N; X4 представляет собой R, P, S, G, L, A или T; X5 представляет собой F, R, Y, V или T; X6 представляет собой L, D, A, S, T, V или отсутствует; X7 представляет собой G, R или отсутствует; X8 представляет собой S, G, V или отсутствует; X9 представляет собой T, A, G, N, S или P; Х10 представляет собой D, Y или E; и X11 представляет собой T или E.
[0298] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vβ, содержащую область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность AX2X3X4X5X6X7X8X91YEQY (SEQ ID NO: 1197), где X2 представляет собой S, M, I, или К; X3 представляет собой S, T, A или N; X4 представляет собой P, S, G, T или A; Х5 представляет собой R или Y; X6 представляет собой D, A, S, T или V; X7 представляет собой R или отсутствует; X8 представляет собой G, V или отсутствует; и X9 представляет собой S, T, A или N.
[0299] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vβ, содержащую область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность ASTX4X5X6X7X8X9X10X11EX13X14 (SEQ ID NO: 1198), где X4 представляет собой T, P или G; Х5 представляет собой R или S; X6 представляет собой S, D, G или V; X7 представляет собой D или отсутствует; X8 представляет собой S или отсутствует; X9 представляет собой S, R или отсутствует; X10 представляет собой S, T, Y или G; X11 представляет собой Y, N или K; X13 представляет собой Q или A; и X14 представляет собой Y или F.
[0300] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vβ, содержащую область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность AX2X3X4X5X6X7X8YGYT (SEQ ID NO: 1199), где X2 представляет собой S или I; Х3 представляет собой S или Т; Х4 представляет собой L, A или D; X5 представляет собой L, T или R; X6 представляет собой L, T или R; X7 представляет собой G, D или отсутствует; и Х8 представляет собой А или N.
[0301] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vβ, содержащую область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность AX2X3X4X5X6X7X8X9X10X11X12X13X14 (SEQ ID NO: 1200), где X2 представляет собой S, V или I; X3 представляет собой S, N или A; X4 представляет собой R, V, S, L, P, G, I или A; X5 представляет собой F, G, Y, L, V, R, T или S; X6 представляет собой L, G, A, D, R, V или отсутствует; X7 представляет собой G, D, R, S, T или отсутствует; X8 представляет собой S или отсутствует; X9 представляет собой S, H, G, V, T, D, L или отсутствует; X10 представляет собой T, S, A, G, P, N или Y; X11 представляет собой D, Y, E, G или N; Х12 представляет собой Т, Е, G или К; X13 представляет собой Q, Y или L; и X14 представляет собой Y, F, T или I.
[0302] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vβ, содержащую область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность ASSX4X5X6X7X8X9X10X11X12X13X14 (SEQ ID NO: 1201), где X4 представляет собой R, V, S, L, G или A; X5 представляет собой F, G, Y, L, V, T или S; X6 представляет собой A, L, R, D, G или отсутствует; X7 представляет собой G, D, T или отсутствует; X8 представляет собой S или отсутствует; X9 представляет собой S, H, G, T, D, L или отсутствует; X10 представляет собой T, S, A, G, P, N или Y; X11 представляет собой D, Y, E, G или N; Х12 представляет собой Т, Е, G или Т; X13 представляет собой Q, Y или L; и X14 представляет собой Y, F или T.
[0303] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vβ, содержащую область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность ASSX4X5X6X7X8X9X10TQY (SEQ ID NO: 1202), где X4 представляет собой R, L или G; X5 представляет собой F, V, T или Y; X6 представляет собой L, A или отсутствует; X7 представляет собой G или отсутствует; X8 представляет собой S, G или отсутствует; X9 представляет собой T, G, P или S; и X10 представляет собой D или E.
[0304] В некоторых вариантах осуществления область Vβ содержит область 1, определяющую комплементарность, (CDR-1), содержащую аминокислотную последовательность SX2X3X4X5 (SEQ ID NO: 1203), где X2 представляет собой G или N; Х3 представляет собой Н или D; Х4 представляет собой Т, L, N или V; и X5 представляет собой A, S, Y или T.
[0305] В некоторых вариантах осуществления область Vβ содержит область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность X1X2X3X4X5X6 (SEQ ID NO: 1204), где X1 представляет собой F или Y; Х2 представляет собой Q, Y или N; X3 представляет собой G, N, R или Y; X4 представляет собой N, G, E или T; X5 представляет собой S, E, A или G; и X6 представляет собой A, E, I или Q.
[0306] В некоторых аспектах область Vβ содержит область 1, определяющую комплементарность, (CDR-1), содержащую аминокислотную последовательность, представленную в SEQ ID NO: 154, 701, 719 или 751. В некоторых вариантах осуществления область Vβ содержит область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность, представленную в SEQ ID NO: 155, 702, 720, 752, 918 или 1009.
[0307] В некоторых вариантах осуществления область Vα содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 153, 159, 301, 694, 712, 729, 744, 762, 776, 788, 802, 818, 832, 846, 858, 870, 882, 896, 911, 926, 940, 952, 964, 976, 988, 1002 или 1391, или CDR3, содержащиеся в аминокислотной последовательности, представленной в любой из SEQ ID NO: 117, 119, 295, 691, 709, 726, 741, 759, 775, 787, 799, 815, 830, 845, 857, 869, 881, 895, 908, 925, 937, 951, 963, 975, 987, 999 или 1390. В некоторых вариантах осуществления область Vα содержит последовательность CDR3, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичную таким последовательностям.
[0308] В некоторых вариантах осуществления область Vα дополнительно содержит область 1, определяющую комплементарность, (CDR-1), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 151, 157, 171, 692, 710, 727, 742, 760, 800, 816, 909, 938 или 1000, или последовательность, имеющую, по меньшей мере или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью. В некоторых аспектах область Vα дополнительно содержит область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 152, 158, 172, 693, 711, 728, 743, 761, 801, 817, 831, 910, 939 или 1001, или последовательность, имеющую, по меньшей мере или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью.
[0309] В некоторых аспектах область Vβ содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 156, 160, 703, 721, 736, 753, 769, 782, 794, 809, 825, 840, 852, 864, 876, 888, 902, 919, 932, 946, 958, 970, 982, 994, 1010 или 1381, или CDR3, содержащуюся в аминокислотной последовательности, представленной в любой из SEQ ID NO: 118, 120, 296, 700, 718, 735, 750, 768, 781, 793, 808, 824, 839, 851, 863, 875, 887, 901, 917, 931, 945, 957, 969, 981 993, 1008, 1380. В некоторых вариантах осуществления область Vβ содержит последовательность CDR3, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99%, идентичную таким последовательностям. В некоторых вариантах осуществления область Vβ содержит область 1, определяющую комплементарность, (CDR-1), содержащую аминокислотную последовательность, представленную в SEQ ID NO: 154, 701, 719 или 751, или последовательность, имеющую по меньшей мере или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью. В некоторых случаях область Vβ содержит область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность, представленную в SEQ ID NO: 155, 702, 720, 752, 918 или 1009, или последовательность, имеющую по меньшей мере или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью.
[0310] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vα, которая содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 151, 152 и 153, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 156, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0311] В некоторых аспектах область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 157, 158 и 159, соответственно. В некоторых таких аспектах область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 160, соответственно. Также среди предложенных TCR имеются те, которые имеют последовательности, по меньшей мере, на 90 или 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичны таким последовательностям.
[0312] В некоторых вариантах осуществления область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 151, 152 и 301, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 156, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0313] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vα, которая содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 692, 693 и 694, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 701, 702 и 703, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0314] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vα, которая содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 710, 711 и 712, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 719, 720 и 721, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0315] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vα, которая содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 727, 728 и 729, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 736, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0316] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vα, которая содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 742, 743 и 744, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 751, 752 и 753, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0317] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vα, которая содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 760, 761 и 762, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 719, 720 и 769, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0318] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vα, которая содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 171, 172 и 776, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 782, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0319] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vα, которая содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 742, 743 и 788, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 139, 140 и 794, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0320] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vα, которая содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 800, 801 и 802, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 751, 752 и 809, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0321] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vα, которая содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 816, 817 и 818, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 825, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0322] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vα, которая содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 816, 831 и 832, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 840, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0323] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vα, которая содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 171, 172 и 846, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 852, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0324] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vα, которая содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 816, 831 и 858, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 864, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0325] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vα, которая содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 727, 728 и 870, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 876, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0326] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vα, которая содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 570, 571 и 882, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 719, 720 и 888, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0327] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vα, которая содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 816, 817 и 896, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 701, 702 и 902, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0328] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vα, которая содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 909, 910 и 911, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 701, 918 и 919, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0329] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vα, которая содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 727, 728 и 926, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 932, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0330] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vα, которая содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 938, 939 и 940, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 946, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0331] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vα, которая содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 727, 728 и 952, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 958, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0332] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vα, которая содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 151, 152 и 964, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 719, 720 и 970, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0333] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vα, которая содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 727, 728 и 976, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 982, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0334] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vα, которая содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 710, 711 и 988, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 719, 720 и 994, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0335] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vα, которая содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 1000, 1001 и 1002, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 139, 1009 и 1010, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0336] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат область Vα, которая содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 171, 172 и 1391, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 1381, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0337] В некоторых случаях область Vα содержит область 1, определяющую комплементарность, (CDR-1), CDR-2 и CDR-3, соответственно содержащие аминокислотные последовательности CDR-1, CDR-2 и CDR-3, содержащиеся в аминокислотной последовательности области Vα, представленной в любой из SEQ ID NO: 117, 119, 295, 691, 709, 726, 741, 759, 775, 787, 799, 815, 830, 845, 857, 869, 881, 895, 908, 925, 937, 951, 963, 975, 987, 999 или 1390. В некоторых случаях область Vβ содержит область 1, определяющую комплементарность, (CDR-1), CDR-2 и CDR-3, соответственно, содержащие аминокислотные последовательности CDR-1, CDR-2 и CDR-3, содержащиеся в аминокислотной последовательности области Vβ, представленной в любой из SEQ ID NO: 118, 120, 296, 700, 718, 735, 750, 768, 781, 793, 808, 824, 839, 851, 863, 875, 887, 901, 917, 931, 945, 957, 969, 981, 993, 1008 или 1380. Также среди представленных TCR имеются такие, которые содержат последовательности, по меньшей мере, или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0338] В некоторых вариантах осуществления TCR или антигенсвязывающий фрагмент включают область Vα, которая содержит область 1, определяющую комплементарность, (CDR-1), CDR-2 и CDR-3, соответственно, содержащие аминокислотные последовательности CDR-1, CDR-2, и CDR-3, представленные в Таблице 5, и область Vβ, которая содержит область 1, определяющую комплементарность, (CDR-1), CDR-2 и CDR-3, соответственно, содержащие аминокислотные последовательности CDR-1, CDR-2, и CDR-3, представленные в Таблице 5. Также среди представленных TCR имеются такие, которые содержат последовательности, по меньшей мере, или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям. Иллюстративные TCR, содержащие такие CDR, или их модифицированные версии, как описано в другом месте настоящего документа, также представлены в Таблице 5.
[0339] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат области Vα и Vβ, содержащие аминокислотные последовательности SEQ ID NO: 117 и 118 или 296, соответственно. В некоторых аспектах области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 119 и 120, соответственно. В некоторых аспектах области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 295 и 118 или 296, соответственно. Также среди представленных TCR имеются такие, которые содержат последовательности, по меньшей мере, или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям. В некоторых случаях области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 691 и 700, соответственно. В некоторых случаях области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 709 и 718, соответственно. В некоторых аспектах области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 726 и 735, соответственно. В некоторых вариантах осуществления области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 741 и 750, соответственно. В некоторых случаях области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 759 и 768, соответственно. В некоторых аспектах области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 775 и 781, соответственно. В некоторых вариантах осуществления области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 787 и 793, соответственно. В некоторых примерах области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 799 и 808, соответственно. В некоторых случаях области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 815 и 824, соответственно. В некоторых случаях области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 830 и 839, соответственно. В некоторых вариантах осуществления области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 845 и 851, соответственно. В некоторых аспектах области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 857 и 863, соответственно. В некоторых случаях области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 869 и 875, соответственно. В некоторых случаях области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 881 и 887, соответственно. В некоторых вариантах осуществления области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 895 и 901, соответственно. В некоторых аспектах области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 908 и 917, соответственно. В некоторых случаях области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 925 и 931, соответственно. В некоторых случаях области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 937 и 945, соответственно. В некоторых примерах области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 951 и 957, соответственно. В некоторых случаях области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 963 и 969, соответственно. В некоторых случаях области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 975 и 981, соответственно. В некоторых случаях области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 987 и 993, соответственно. В некоторых вариантах осуществления области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 999 и 1008, соответственно. В некоторых вариантах осуществления области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 1390 и 1380, соответственно.
[0340] В некоторых вариантах осуществления альфа-цепь TCR или его антигенсвязывающий фрагмент дополнительно содержат область Cα или ее часть, и/или бета-цепь дополнительно содержит область Cβ или ее часть. В некоторых вариантах осуществления область Cα или ее часть содержат аминокислотную последовательность, представленную в любой из SEQ ID NO: 213, 217, 218 или 524, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности, такая как последовательность, имеющая по меньшей мере или примерно 90, 91, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью. В некоторых аспектах область Cβ содержит аминокислотную последовательность, представленную в SEQ ID NO: 214, 216, 631 или 889, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности, такую как последовательность, имеющая в по меньшей мере или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью. В некоторых вариантах осуществления области Cα и/или Cβ модифицируют, например, путем включения одного или более ненативных остатков цистеина, таких как любые, описанные в настоящем документе. В некоторых вариантах осуществления область Cα или ее часть содержат ненативный цистеин в остатке 48 и содержит аминокислотную последовательность, представленную в любой из SEQ ID NO: 196, 200, 201, 203 или 525, или последовательность аминокислот, которая имеет с ними, по меньшей мере, 90% идентичности последовательности, такую как последовательность, имеющую, по меньшей мере, или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью и которая содержит введенный ненативный цистеиновый остаток (например, Cys48). В некоторых аспектах область Cβ содержит ненативный цистеин в остатке 57 и содержит аминокислотную последовательность, представленную в SEQ ID NO: 197, 199, 890 или 1363, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности, такая как последовательность, имеющая по меньшей мере или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью.
[0341] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат альфа-цепь, содержащую последовательность аминокислот, представленную в SEQ ID NO: 48, 58, 283, 687, 705, 722, 737, 755, 771, 783, 795, 811, 826, 841, 853, 865, 877, 891, 904, 921, 933, 947, 959, 971, 983, 995 или 1386, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности, такая как последовательность, имеющая, по меньшей мере, или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью, и/или бета-цепь, содержащую последовательность аминокислот, представленную в SEQ ID NO: 52, 285, 62, 696, 714, 731, 746, 764, 777, 789, 804, 820, 835, 847, 859, 871, 883, 897, 913, 927, 941, 953, 965, 977, 989, 1004 или 1376, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности, такая как последовательность, имеющая по меньшей мере или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью.
[0342] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат альфа-цепь, содержащую последовательность аминокислот, представленную в SEQ ID NO: 49, 59, 284, 688, 706, 723, 738, 756, 772, 784, 796, 812, 827, 842, 854, 866, 878, 892, 905, 922, 934, 948, 960, 972, 984, 996 или 1387, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности, такая как последовательность, имеющая, по меньшей мере, или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью, и/или бета-цепь, содержащая последовательность аминокислот, представленную в SEQ ID NO: 53, 63, 286, 697, 715, 732, 747, 765, 778, 790, 805, 821, 836, 848, 860, 872, 884, 898, 914, 928, 928, 942, 954, 966, 978, 990, 1005 или 1377, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности, такая как последовательность, имеющая по меньшей мере или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью.
[0343] В некоторых вариантах осуществления области Vα и Vβ содержат аминокислотные последовательности, соответствующие SEQ ID NO, представленным в Таблице 6 или Таблице 7. В некоторых аспектах TCR содержит последовательности константной области альфа и константной области бета, такие как последовательности, соответствующие SEQ ID NO, представленным в Таблице 6 или Таблице 7. В некоторых случаях TCR содержит полную последовательность, содержащую вариабельную и константную области цепи, такую как последовательность, соответствующая SEQ ID NO, представленным в Таблице 6 или Таблице 7 («Полная»). В некоторых вариантах осуществления полная последовательность, содержащая вариабельную и константную области, также включает сигнальную последовательность и, таким образом, содержит последовательность, соответствующую SEQ ID NO, представленным в Таблице 6 или Таблице 7 («Полная+сигнальная»). Также среди представленных TCR имеются такие, которые содержат последовательности, по меньшей мере, или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям. Иллюстративные TCR, содержащие такие последовательности, или их модифицированные версии, как описано в другом месте настоящего документа, также представлены в Таблицах 6 и 7, соответственно.
(Vβ)
c. HPV 16 Е7 (86-93)
[0344] В некоторых случаях TCR распознает или связывает пептидный эпитоп, полученный из E7 HPV 16, который представляет собой или содержит E7 (86-93) TLGIVCPI (SEQ ID NO: 235). В некоторых вариантах осуществления TCR распознает или связывает E7 HPV 16 (86-93) в контексте MHC, такого как MHC класса I, например, HLA-A2.
[0345] В некоторых вариантах осуществления область Vα содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность, представленную в SEQ ID NO: 175. В некоторых вариантах осуществления область Vα содержит последовательность CDR3, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичную таким последовательностям. В некоторых аспектах область Vα содержит область 1, определяющую комплементарность, (CDR-1), содержащую аминокислотную последовательность, представленную в SEQ ID NO: 142, или последовательность, имеющую по меньшей мере или примерно 90, 91, 92, 93, 94 95, 96, 97, 98 или 99% идентичности с такой последовательностью. В некоторых аспектах область Vα содержит область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность, представленную в SEQ ID NO: 143, или последовательность, имеющую по меньшей мере или примерно 90, 91, 92, 93, 94 95, 96, 97, 98 или 99% идентичности с такой последовательностью.
[0346] В некоторых вариантах осуществления область Vβ содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность, представленную в SEQ ID NO: 178, или последовательность, имеющую по меньшей мере или примерно 90, 91, 92, 93, 94 95, 96, 97, 98 или 99% идентичности с такой последовательностью. В некоторых случаях область Vβ содержит область 1, определяющую комплементарность, (CDR-1), содержащую аминокислотную последовательность, представленную в SEQ ID NO: 176, или последовательность, имеющую по меньшей мере или примерно 90, 91, 92, 93, 94 95, 96, 97, 98 или 99% идентичности с такой последовательностью. В некоторых аспектах область Vβ содержит область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность, представленную в SEQ ID NO: 177, или последовательность, имеющую по меньшей мере или примерно 90, 91, 92, 93, 94 95, 96, 97, 98 или 99% идентичности с такой последовательностью.
[0347] В некоторых вариантах осуществления область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 142, 143 и 175, соответственно. В некоторых таких вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 176, 177 и 178, соответственно. Также среди представленных TCR имеются такие, которые имеют последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0348] В некоторых аспектах область Vα содержит область 1, определяющую комплементарность, (CDR-1), CDR-2 и CDR-3, соответственно, содержащие аминокислотные последовательности CDR-1, CDR-2 и CDR-3, содержащиеся в аминокислотной последовательности области Vα, представленной в SEQ ID NO: 127. В некоторых вариантах осуществления область Vβ содержит CDR-1, CDR-2 и CDR-3, соответственно, содержащие аминокислотные последовательности CDR-1, CDR-2 и CDR-3, содержащиеся в аминокислотной последовательности области Vβ, представленной в SEQ ID NO: 128. Также среди представленных TCR имеются такие, которые содержат последовательности, по меньшей мере, или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0349] В некоторых вариантах осуществления TCR или антигенсвязывающий фрагмент включают область Vα, содержащую область 1, определяющую комплементарность, (CDR-1), CDR-2 и CDR-3, соответственно содержащие аминокислоные последовательности CDR-1, CDR-2 и CDR-3, представленные в Таблице 8, и область Vβ, содержащую область 1, определяющую комплементарность, (CDR-1), CDR-2 и CDR-3, соответственно, содержащие аминокислотные последовательности CDR-1, CDR-2 и CDR-3, представленные в Таблице 8. Также среди представленных TCR имеются такие, которые содержат последовательности, по меньшей мере, или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям. Иллюстративные TCR, содержащие такие CDR, или их модифицированные версии, как описано в другом месте настоящего документа, также представлены в Таблице 8.
[0350] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат области Vα и Vβ, содержащие аминокислотные последовательности SEQ ID NO: 127 и 128, соответственно. Также среди представленных TCR имеются такие, которые содержат последовательности, по меньшей мере, или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям.
[0351] В некоторых вариантах осуществления альфа-цепь TCR или его антигенсвязывающий фрагмент дополнительно содержат область Cα или ее часть, и/или бета-цепь дополнительно содержит область Cβ или ее часть. В некоторых вариантах осуществления область Cα или ее часть содержит аминокислотную последовательность, представленную в любой из SEQ ID NO: 212, 213 или 217, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности, такую как последовательность, имеющая по меньшей мере или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью. В некоторых аспектах область Cβ содержит аминокислотную последовательность, представленную в SEQ ID NO: 214 или 216, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности, такую как последовательность, имеющая, по меньшей мере, или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью. В некоторых вариантах осуществления области Cα и/или Cβ модифицируют, например, путем включения одного или более ненативных остатков цистеина, таких как любые, описанные в настоящем документе. В некоторых вариантах осуществления область Cα или ее часть содержит ненативный цистеин в остатке 48 и содержит аминокислотную последовательность, представленную в SEQ ID NO: 200, или последовательность аминокислот, которая имеет с ней по меньшей мере 90% идентичность последовательности, такую как последовательность, имеющая, по меньшей мере или примерно 90, 91, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью и которая содержит введенный ненативный цистеиновый остаток (например, Cys48). В некоторых аспектах область Cβ содержит ненативный цистеин в остатке 57 и содержит аминокислотную последовательность, представленную в SEQ ID NO: 197 или 199, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности, такую как последовательность, имеющая по меньшей мере или примерно 90, 91, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью.
[0352] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат альфа-цепь, содержащую последовательность аминокислот, представленную в SEQ ID NO: 98, или последовательность аминокислот, которая имеет с ней по меньшей мере 90% идентичности последовательности, такую как последовательность, имеющая, по меньшей мере, или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью, и/или бета-цепь, содержащую последовательность аминокислот, представленную в SEQ ID NO: 102, или последовательность аминокислот, которая имеет с ней по меньшей мере 90% идентичности, такую как последовательность, имеющая по меньшей мере или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью.
[0353] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент содержат альфа-цепь, содержащую последовательность аминокислот, представленную в SEQ ID NO: 99, или последовательность аминокислот, которая имеет с ней по меньшей мере 90% идентичности последовательности, такую как последовательность, имеющая, по меньшей мере, или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью, и/или бета-цепь, содержащая последовательность аминокислот, представленную в SEQ ID NO: 103, или последовательность аминокислот, которая имеет с ней по меньшей мере 90% идентичности, такую как последовательность, имеющая по меньшей мере или примерно 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичности с такой последовательностью.
[0354] В некоторых вариантах осуществления области Vα и Vβ содержат аминокислотные последовательности, соответствующие SEQ ID NO, представленным в Таблице 9 или Таблице 10. В некоторых аспектах TCR содержит последовательности константной области альфа и константной области бета, такие как последовательности, соответствующие SEQ ID NO, представленным в Таблице 9 или Таблице 10. В некоторых случаях TCR содержит полную последовательность, содержащую вариабельную и константную области цепи, такую как последовательность, соответствующая SEQ ID NO, представленным в Таблице 9 или Таблице 10 («Полная»). В некоторых вариантах осуществления полная последовательность, содержащая вариабельную и константную области, также включает сигнальную последовательность и, таким образом, содержит последовательность, соответствующую SEQ ID NO, представленным в Таблице 9 или Таблице 10 («Полныйая+сигнальная»). Также среди представленных TCR имеются такие, которые содержат последовательности, по меньшей мере, или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99% идентичные таким последовательностям. Иллюстративные TCR, содержащие такие последовательности, или их модифицированные версии, как описано в другом месте настоящего документа, также представлены в Таблицах 9 и 10, соответственно.
2. Варианты и модификации
[0355] В некоторых вариантах осуществления связывающая молекула, например TCR или его антигенсвязывающий фрагмент, являются модифицированными или были модифицированы. В некоторых вариантах осуществления связывающие молекулы, например TCR или их антигенсвязывающие фрагменты, включают одну или более вариаций аминокислот, например замен, делеций, вставок и/или мутаций, по сравнению с последовательностью связывающей молекулы, например, TCR, описанной в настоящем документе. Иллюстративные варианты включают варианты, предназначенные для улучшения аффинности связывания и/или других биологических свойств молекулы связывания. Варианты аминокислотных последовательностей связывающей молекулы могут быть получены путем введения соответствующих модификаций в нуклеотидную последовательность, кодирующую связывающую молекулу, или путем пептидного синтеза. Такие модификации включают, например, делеции и/или вставки и/или замены остатков в аминокислотных последовательностях связывающей молекулы. Любая комбинация делеции, вставки и замены может быть сделана для достижения конечной конструкции, при условии, что конечная конструкция обладает желаемыми характеристиками, например, связыванием антигена.
[0356] В некоторых вариантах осуществления способы направленной эволюции используются для создания TCR с измененными свойствами, такими как более высокая аффинность к конкретному пептиду в контексте молекулы МНС. В некоторых вариантах осуществления направленная эволюция достигается методами дисплея, включая, но не ограничиваясь этим, дрожжевой дисплей (Holler et al. (2003) Nat Immunol, 4, 55-62; Holler et al. (2000) Proc Natl Acad Sci USA, 97, 5387-92), фаговый дисплей (Li et al. (2005) Nat Biotechnol, 23, 349-54) или Т-клеточный дисплей (Chervin et al. (2008) J Immunol Methods, 339, 175-84). В некоторых вариантах осуществления способы дисплея включают конструирование или модификацию известного, родительского или эталонного TCR. Например, в некоторых случаях эталонный TCR, такой как любой, представленный в настоящем описании, может использоваться в качестве матрицы для получения мутагенизированных TCR, в которых в одном или более остатках CDR мутированы, и выбираются мутанты с желаемым измененным свойством, такими как более высокая аффмнность к пептидному эпитопу в контексте молекулы МНС.
[0357] В некоторых вариантах осуществления связывающие молекулы, например TCR или их антигенсвязывающие фрагменты, включают одну или более аминокислотных замен, например, по сравнению с последовательностью связывающей молекулой, например, TCR, описанной в настоящем документе, и/или по сравнению с последовательностью естественного репертуара, например, человеческого репертуара. Сайты, представляющие интерес для заместительного мутагенеза, включают области CDR, FR и/или константные области. Аминокислотные замены могут быть введены в представляющую интерес связывающую молекулу, и продукты подвергаются скринингу на предмет желаемой активности, например, сохраненной/улучшенной антигенной аффинности или авидности, сниженной иммуногенности, улучшенного периода полужизни, CD8-независимого связывания или активности, поверхностной экспрессии, стимулирования спаривания цепей TCR и/или других улучшенных свойств или функций.
[0358] В некоторых вариантах осуществления один или более остатков в CDR родительской связывающей молекулы, например TCR, является/являются замещенными. В некоторых вариантах осуществления замена выполняется для возвращения последовательности или положения в последовательности к последовательности зародышевой линии, такой как последовательность связывающей молекулы, присутствующая в зародышевой линии (например, человеческой зародышевой линии), например, для уменьшения вероятности иммуногенности, например, после введения человеку.
[0359] В некоторых вариантах осуществления замены, вставки или делеции могут происходить в одной или более CDR, если такие изменения существенно не снижают способность связывающей молекулы, например TCR или ее антигенсвязывающего фрагмента, связывать антиген. Например, в CDR могут быть сделаны консервативные изменения (например, консервативные замены, как предусмотрено в настоящем документе), которые существенно не снижают аффинность связывания. Такие изменения могут, например, присутствовать вне антигенсвязывающих остатков в CDR. В определенных вариантах осуществления вариабельных последовательностей, предложенных в настоящем описании, каждая CDR либо не изменяется, либо содержит не более одной, двух или трех аминокислотных замен.
[0360] Вставки аминокислотной последовательности включают амино- и/или карбоксиконцевые слияния длиной от одного остатка до полипептидов, содержащих сто или более остатков, а также вставки внутри последовательности одного или более аминокислотных остатков.
[0361] В некоторых аспектах TCR или его антигенсвязывающий фрагмент может содержать одну или более модификаций в альфа-цепи и/или бета-цепи, так что когда TCR или его антигенсвязывающий фрагмент экспрессируются в клетке, частота ошибочного спаривания между альфа-цепью и бета-цепью TCR и эндогенной альфа-цепью и бета-цепью TCR снижается, экспрессия альфа-цепи и бета-цепи TCR увеличивается и/или повышается стабильность альфа-цепи и бета-цепи TCR.
[0362] В некоторых вариантах осуществления TCR содержит один или более ненативных остатков цистеина для введения ковалентной дисульфидной связи, связывающей остаток области иммуноглобулина константного домена α-цепи с остатком области иммуноглобулина константного домена β-цепи. В некоторых вариантах осуществления один или более цистеинов могут быть включены во внеклеточные последовательности константной области первого и второго сегментов полипептида TCR. Иллюстративные неограничивающие модификации в TCR для введения ненативных остатков цистеина описаны в настоящем документе (см. также международную публикацию PCT No. WO2006/000830 и WO2006037960). В некоторых случаях может быть желательной как нативная, так и ненативная дисульфидная связь. В некоторых вариантах осуществления TCR или антигенсвязывающий фрагмент модифицируют таким образом, чтобы в нативном TCR отсутствовала межцепочечная дисульфидная связь.
[0363] В некоторых вариантах осуществления трансмембранный домен константной области TCR может быть модифицирован так, чтобы он содержал большее количество гидрофобных остатков (см., например, Haga-Friedman et al. (2012) Journal of Immunology, 188: 5538-5546). В некоторых вариантах осуществления трансмембранная область α-цепи TCR содержит одну или более мутаций, соответствующих S116L, G119V или F120L, применительно к нумерации Cα, представленной в любой из SEQ ID NO: 212, 213, 215, 217, 220, или 524.
[0364] В некоторых вариантах осуществления клетка, экспрессирующая TCR, дополнительно включает маркер, такой как маркер клеточной поверхности, который может использоваться для подтверждения трансдукции или конструирования клетки для экспрессии TCR, такого как укороченная версия рецептора клеточной поверхности, такого как укороченный EGFR (tEGFR). Иллюстративные суррогатные маркеры могут включать укороченные формы полипептидов клеточной поверхности, такие как укороченные формы, которые не функциональны и не трансдуцируют или не способны к трансдукции сигнала, или сигнала, обычно трансдуцируемого полноразмерной формой полипептида клеточной поверхности, и/или не интернализуются или не способны интернализоваться. Иллюстративные укороченные полипептиды клеточной поверхности, включая укороченные формы факторов роста или других рецепторов, такие как укороченный рецептор 2 эпидермального фактора роста человека (tHER2), укороченный рецептор эпидермального фактора роста (tEGFR, иллюстративная последовательность tEGFR, представленная в SEQ ID NO: 273 или 343) или простат-специфический мембранный антиген (PSMA) или его модифицированная форма. tEGFR может содержать эпитоп, распознаваемый антителом цетуксимаб (Erbitux®) или другим терапевтическим антителом против EGFR или связывающей молекулой, которые можно использовать для идентификации или отбора клеток, которые были сконструированы с помощью конструкции tEGFR и кодируемого экзогенного белка, и/или для устранения или отделения клеток, экспрессирующих кодируемый экзогенный белок. См. Патент США № 8802374 и Liu et al., Nature Biotech. 2016 April; 34 (4): 430-434). В некоторых аспектах маркер, например суррогатный маркер включает весь или часть (например, укороченную форму) CD34, NGFR, CD19 или укороченный CD19, например укороченный CD19, не являющийся человеческим, или рецептор эпидермального фактора роста (например, tEGFR). В некоторых вариантах осуществления маркер представляет собой или содержит флуоресцентный белок, такой как зеленый флуоресцентный белок (GFP), усиленный зеленый флуоресцентный белок (EGFP), такой как супер-кратный GFP (sfGFP), красный флуоресцентный белок (RFP), такой как tdTomato, mCherry, mStrawberry, AsRed2, DsRed или DsRed2, голубой флуоресцентный белок (CFP), сине-зеленый флуоресцентный белок (BFP), усиленный синий флуоресцентный белок (EBFP) и желтый флуоресцентный белок (YFP) и их варианты, включая видовые варианты, мономерные варианты и кодон-оптимизированные и/или улучшенные варианты флуоресцентных белков. В некоторых вариантах осуществления маркер представляет собой или содержит фермент, такой как люцифераза, ген lacZ из E. coli, щелочную фосфатазу, секретируемую эмбриональную щелочную фосфатазу (SEAP), хлорамфениколацетилтрансферазу (CAT). Иллюстративные светоизлучающие репортерные гены включают люциферазу (luc), β-галактозидазу, хлорамфениколацетилтрансферазу (CAT), β-глюкуронидазу (GUS) или их варианты.
[0365] В некоторых вариантах осуществления маркер является маркером селекции. В некоторых вариантах осуществления маркер селекции представляет собой или содержит полипептид, который придает устойчивость к экзогенным агентам или лекарственным средствам. В некоторых вариантах осуществления маркер селекции представляет собой ген устойчивости к антибиотику. В некоторых вариантах осуществления маркер селекции представляет собой ген устойчивости к антибиотику, придающий устойчивость к антибиотику клетке млекопитающего. В некоторых вариантах осуществления маркер селекции представляет собой или содержит ген устойчивости к пуромицину, ген устойчивости к гигромицину, ген устойчивости к бластидину, ген устойчивости к неомицину, ген устойчивости к генетицину или ген устойчивости к зеоцину или их модифицированную форму.
[0366] В некоторых аспектах маркер включает весь или часть (например, укороченную форму) CD34, NGFR или рецептора эпидермального фактора роста (например, tEGFR). В некоторых вариантах осуществления нуклеиновая кислота, кодирующая маркер, функционально связана с полинуклеотидом, кодирующим последовательность линкера, такую как расщепляемая последовательность линкера, например, T2A. Смотри WO2014031687. В некоторых вариантах осуществления введение конструкции, кодирующей TCR и EGFRt, разделенных T2A, P2A или другим переключателем рибосомы, может позволить экспрессировать два белка из одной и той же конструкции, так что EGFRt можно использовать в качестве маркера для детектирования клеток, экспрессирующих такую конструкцию. Примеры таких маркеров, которые можно использовать, описаны ниже.
[0367] В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент кодируется нуклеотидной последовательностью, которая оптимизирована или была оптимизирована по кодонам. Иллюстративные кодон-оптимизированные варианты описаны в другом месте настоящего документа.
B. Антитела
[0368] В некоторых вариантах осуществления связывающая молекула представляет собой антитело или его антигенсвязывающий фрагмент, который содержит любую одну или более CDR, как описано выше в отношении TCR.
[0369] В некоторых вариантах осуществления антитело или его антигенсвязывающий фрагмент содержат вариабельную область тяжелой и легкой цепи, содержащие CDR1, CDR2 и/или CDR3, содержащиеся в альфа-цепи, и CDR1, CDR2 и/или CDR3, содержащиеся в бета-цепи, как представлено в Таблице 2, Таблице 5 или Таблице 8. Также среди представленных антител или антигенсвязывающих фрагментов имеются такие, которые содержат последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99%, идентичные таким последовательностям.
[0370] В некоторых вариантах осуществления антитело или антигенсвязывающий фрагмент содержат вариабельную область, которая содержит область 1, определяющую комплементарность, (CDR-1), CDR-2 и CDR-3, соответственно, содержащие аминокислотные последовательности CDR-1, CDR-2, и CDR-3, содержащиеся в аминокислотной последовательности области Vα, представленной в любой из SEQ ID NO: 111, 113, 115, 121, 123 125, 297, 299, 477, 492, 504, 510, 522, 536, 554, 569, 587, 599, 611, 623, 637, 649, 661 или 676. В некоторых аспектах антитело или его антигенсвязывающий фрагмент содержат вариабельную область, которая содержит область 1, определяющую комплементарность, (CDR-1), CDR-2 и CDR-3, соответственно, содержащие аминокислотные последовательности CDR-1, CDR-2, и CDR-3, содержащиеся в аминокислотной последовательности области Vβ, представленной в любой из SEQ ID NO: 112, 114, 116, 122, 124 126, 298, 300, 483, 498, 516, 530, 545, 560, 578, 593, 605, 617, 629, 643, 655, 667 или 685. Также среди представленных антител или антигенсвязывающих фрагментов имеются такие, которые содержат последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99%, идентичные таким последовательностям.
[0371] В некоторых вариантах осуществления представленное антитело или фрагмент антитела представляет собой антитело человека. В некоторых вариантах осуществления представленное антитело или фрагмент антитела содержат область VH, которая содержит часть, имеющую по меньшей мере 95%, 96%, 97%, 98%, 99% или 100% идентичности последовательности с аминокислотной последовательностью, кодируемой нуклеотидной последовательностью V-сегмента тяжелой цепи зародышевой линии человека, часть, которая имеет по меньшей мере 95%, 96%, 97%, 98%, 99% или 100% идентичности с аминокислотной последовательностью, кодируемой нуклеотидной последовательностью D-сегмента тяжелой цепи зародышевой линии человека, и/или часть, имеющую, по меньшей мере, 95%, 96%, 97%, 98%, 99% или 100% идентичности с аминокислотной последовательностью, кодируемой нуклеотидной последовательностью J-сегмента тяжелой цепи зародышевой линии человека; и/или содержит область VL, которая содержит часть, имеющую по меньшей мере 95%, 96%, 97%, 98%, 99% или 100% идентичности последовательности с аминокислотной последовательностью, кодируемой нуклеотидной последовательностью V-сегмента каппа- или лямбда-цепи зародышевой линии человека и/или часть, имеющую по меньшей мере 95%, 96%, 97%, 98%, 99% или 100% идентичности с аминокислотной последовательностью, кодируемой нуклеотидной последовательностью J-сегмента каппа- или лямбда-цепи зародышевой линии человека. В некоторых вариантах осуществления часть области VH соответствует CDR-H1, CDR-H2 и/или CDR-H3. В некоторых вариантах осуществления часть области VH соответствует каркасной области 1 (FR1), FR2, FR2 и/или FR4. В некоторых вариантах осуществления часть области VL соответствует CDR-L1, CDR-L2 и/или CDR-L3. В некоторых вариантах осуществления часть области VL соответствует FR1, FR2, FR2 и/или FR4.
[0372] В некоторых вариантах осуществления антитело или его антигенсвязывающий фрагмент содержат каркасную область, которая содержит последовательности генных сегментов зародышевой линии человека. Например, в некоторых вариантах осуществления антитело или антигенсвязывающий фрагмент содержат область VH, в которой находится каркасная область, например FR1, FR2, FR3 и FR4, имеет по меньшей мере 95%, 96%, 97%, 98%, 99% или 100% идентичности последовательности с каркасной областью, кодируемой сегментом антитела зародышевой линии человека, таким как V- и/или J-сегмент. В некоторых вариантах осуществления человеческое антитело содержит область VL, в которой каркасная область, например FR1, FR2, FR3 и FR4 имеют по меньшей мере 95%, 96%, 97%, 98%, 99% или 100% идентичность последовательности с каркасной областью, кодируемой сегментом антитела зародышевой линии человека, таким как V- и/или сегмент. Например, в некоторых таких вариантах осуществления каркасная последовательность последовательности VH и/или VL отличается не более чем на 10 аминокислот, например не более чем на 9, 8, 7, 6, 5, 4, 3, 2 или 1 аминокислоту по сравнению с каркасной областью, кодируемой сегментом антитела зародышевой линии человека. В некоторых вариантах осуществления антитела и их антигенсвязывающие фрагменты, например TCR-подобные антитела специфически распознают пептидный эпитоп в контексте молекулы MHC, такой как MHC класса I. В некоторых случаях молекула MHC класса I представляет собой молекулу HLA-A2, например, HLA-A2*01.
[0373] В некоторых вариантах осуществления антитело или его антигенсвязывающий фрагмент распознают или связываются с эпитопом или областью E6 HPV16, таким как пептидный эпитоп, содержащий аминокислотную последовательность, представленную в любой из SEQ ID NO: 232-234. В некоторых случаях TCR или его антигенсвязывающий фрагмент, которые распознают или связываются с пептидным эпитопом, полученным из E6 HPV16, представляют собой или содержат последовательность, представленную в SEQ ID NO: 233.
[0374] В некоторых аспектах TCR или антигенсвязывающий фрагмент распознают или связываются с эпитопом или областью белка Е7 HPV16, таким как пептидный эпитоп, содержащий аминокислотную последовательность, представленную в любой из SEQ ID NO: 235-239. В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент не распознает или не связывается с эпитопом E7 (11-19), содержащим аминокислотную последовательность YMLDLQPET (SEQ ID NO: 236). В некоторых случаях пептид, полученный из E7 HPV16, представляет собой или содержит последовательность, представленную в SEQ ID NO: 235.
[0375] Таким образом, в некоторых вариантах осуществления представлены антитела против HPV, включая функциональные фрагменты антител. В некоторых вариантах осуществления домены антител VH и/или VL или их антигенсвязывающий сайт способны специфически связываться с пептидным эпитопом HPV 16. В некоторых вариантах осуществления антитела включают вариабельную область тяжелой цепи и вариабельную область легкой цепи, такие как scFv. Антитела включают антитела, которые специфически связываются с HPV, например E6 HPV 16 или E7 HPV 16. [0382] Среди представленных антител против HPV есть человеческие антитела. Антитела включают выделенные антитела. Также представлены молекулы, содержащие такие антитела, например одноцепочечные белки, слитые белки и/или рекомбинантные рецепторы, такие как химерные рецепторы, включая антигенные рецепторы.
[0376] Термин «антитело» в настоящем описании используется в самом широком смысле и включает поликлональные и моноклональные антитела, включая интактные антитела и функциональные (антигенсвязывающие) фрагменты антител, включая фрагменты (Fab) антигенсвязывающих фрагментов, F(ab')2-фрагменты, Fab-фрагменты, Fv-фрагменты, фрагменты рекомбинантного IgG (rIgG), вариабельные области тяжелой цепи (VH), способные специфически связываться с антигеном, фрагменты одноцепочечных антител, включая одноцепочечные вариабельные фрагменты (scFv), и фрагменты однодоменных антител (например, sdAb, sdFv, нанотело). Термин охватывает генетически сконструированные и/или иным образом модифицированные формы иммуноглобулинов, такие как интратела, пептидные антитела, химерные антитела, полностью человеческие антитела, гуманизированные антитела и гетероконъюгатные антитела, полиспецифичные, например, биспецифичные, антитела, диатела, триатела и тетратела, тандемные ди-scFv, тандемные три-scFv. Если не указано иное, термин «антитело» следует понимать как охватывающий его функциональные фрагменты. Термин также охватывает интактные или полноразмерные антитела, включая антитела любого класса или подкласса, включая IgG и его подклассы, IgM, IgE, IgA и IgD.
[0377] В некоторых вариантах осуществления тяжелая и легкая цепи антитела могут быть полноразмерными или могут представлять собой антигенсвязывающую часть (Fab, F(ab')2, Fv или одноцепочечный фрагмент Fv (scFv)). В других вариантах осуществления константная область тяжелой цепи антитела выбрана, например, из IgG1, IgG2, IgG3, IgG4, IgM, IgA1, IgA2, IgD и IgE, в частности, выбрана, например, из IgG1, IgG2, IgG3 и IgG4, более конкретно, IgG1 (например, из человеческого IgG1). В другом варианте осуществления константная область легкой цепи антитела выбрана, например, из каппа или лямбда, в частности, каппа.
[0378] Среди представленных антител есть фрагменты антител. «Фрагмент антитела» относится к молекуле, отличной от интактного антитела, которая содержит часть интактного антитела, которая связывается с антигеном, с которым связывается интактное антитело. Примеры фрагментов антител включают, но не ограничиваются ими, Fv, Fab, Fab ', Fab'-SH, F(ab')2; диатела; линейные антитела; вариабельные области тяжелой цепи (VH), молекулы одноцепочечных антител, такие как scFvs и однодоменные антитела VH; и полиспецифичные антитела, образованные из фрагментов антител. В конкретных вариантах осуществления антитела представляют собой фрагменты одноцепочечных антител, содержащие вариабельную область тяжелой цепи и/или вариабельную область легкой цепи, такие как scFv.
[0379] Термин «вариабельная область» или «вариабельный домен» при использовании в отношении антитела, такого как фрагмент антитела, относится к домену тяжелой или легкой цепи антитела, который участвует в связывании антитела с антигеном. Вариабельные домены тяжелой цепи и легкой цепи (VH и VL, соответственно) нативного антитела, как правило, имеют сходные структуры, причем каждый домен содержит четыре консервативных каркасных области (FR) и три CDR. (См., например, Kindt et al. Kuby Immunology, 6th ed., W.H. Freeman and Co., стр. 91 (2007). Одного домена VH или VL может быть достаточно для придания антигенсвязывающей специфичности. Кроме того, антитела, которые связываются с конкретным антигеном, могут быть выделены с использованием домена VH или VL из антитела, которое связывается с антигеном, для скрининга библиотеки комплементарных доменов VL или VH, соответственно. Смотри, например, Portolano et al., J. Immunol. 150: 880-887 (1993); Clarkson et al., Nature 352: 624-628 (1991).
[0380] Однодоменные антитела представляют собой фрагменты антител, включающие весь или часть вариабельного домена тяжелой цепи или весь или часть вариабельного домена легкой цепи антитела. В некоторых вариантах осуществления однодоменное антитело представляет собой человеческое однодоменное антитело.
[0381] Фрагменты антител могут быть получены различными способами, включая, но не ограничиваясь этим, протеолитическое расщепление интактного антитела, а также продукцию рекомбинантными клетками-хозяевами. В некоторых вариантах осуществления антитела представляют собой полученные рекомбинантным путем фрагменты, такие как фрагменты, содержащие струкутры, которые не встречаются в природе, такие как фрагменты с двумя или более областями или цепями антител, соединенными синтетическими линкерами, например пептидными линкерами, и/или которые могут не получаться путем ферментативного расщепления природного интактного антитела. В некоторых аспектах фрагменты антител представляют собой scFv.
[0382] Среди представленных антител против HPV есть человеческие антитела. «Антитело человека» представляет собой антитело с аминокислотной последовательностью, соответствующей последовательности антитела, продуцируемого человеком или клеткой человека, или источником, не являющимся человеческим, который использует репертуар антител человека или другие последовательности, кодирующие антитела человека, включая библиотеки антител человека. Термин исключает гуманизированные формы антител, не являющихся человеческими, содержащих антигенсвязывающие области, не являющиеся человеческими, такие как те, в которых все или по существу все CDR не являются человеческими. Термин включает антигенсвязывающие фрагменты антител человека.
[0383] «Гуманизированное» антитело представляет собой антитело, в котором все или по существу все аминокислотные остатки CDR получены из CDR, не являющихся человеческими, и все или по существу все аминокислотные остатки FR получены из человеческих FR. Гуманизированное антитело необязательно может включать по меньшей мере часть константной области антитела, полученной из человеческого антитела. «Гуманизированная форма» антитела, не являющегося человеческим относится к варианту антитела, не являющегося человеческим, которое подверглось гуманизации, как правило, для снижения иммуногенности у человека, сохраняя при этом специфичность и аффинность родительского антитела, не являющегося человеческим. В некоторых вариантах осуществления некоторые остатки FR в гуманизированном антителе замещены соответствующими остатками антитела, не являющегося человеческим (например, антитела, из которого получены остатки CDR), например, для восстановления или улучшения специфичности или аффинности антитела.
[0384] Человеческие антитела могут быть получены введением иммуногена трансгенному животному, которое было модифицировано для производства интактных человеческих антител или интактных антител с человеческими вариабельными областями в ответ на антигенную стимуляцию. Такие животные обычно содержат все или часть локусов иммуноглобулина человека, которые заменяют эндогенные локусы иммуноглобулина или которые присутствуют вне хромосом или случайным образом интегрированы в хромосомы животного. У таких трансгенных животных эндогенные локусы иммуноглобулина обычно инактивированы. Антитела человека также могут быть получены из библиотек антител человека, включая фаговый дисплей и бесклеточные библиотеки, содержащие кодирующие антитела последовательности, полученные из репертуара человека.
[0385] В число представленных антител входят моноклональные антитела, включая фрагменты моноклональных антител. Используемый в настоящем описании термин «моноклональное антитело» относится к антителу, полученному из популяции или внутри популяции по существу гомогенных антител, т.е. индивидуальные антитела, составляющие популяцию, являются идентичными, за исключением возможных вариантов, содержащих природные мутации или возникающие во время продуцирования препарата моноклонального антитела, причем такие варианты, как правило, присутствуют в незначительных количествах. В отличие от препаратов поликлональных антител, которые обычно включают разные антитела, направленные против разных эпитопов, каждое моноклональное антитело препарата моноклональных антител направлено против одного эпитопа на антигене. Термин не следует истолковывать как требующий получения антитела каким-либо конкретным способом. Моноклональное антитело может быть получено различными способами, включая, но не ограничиваясь этим, получение из гибридомы, методы рекомбинантной ДНК, фаговый дисплей и другие методы дисплея антител.
[0386] Используемый в настоящем описании термин «соответствующая форма» антитела означает, что при сравнении свойства или активности двух антител это свойство сравнивается с использованием одной и той же формы антитела. Например, если указано, что антитело обладает большей активностью по сравнению с активностью соответствующей формы первого антитела, это означает, что конкретная форма, такая как scFv этого антитела, обладает большей активностью по сравнению с формой scFv первого антитела.
[0387] «Эффекторные функции» относятся к тем биологическим активностям, которые относятся к области Fc антитела, которые зависят от изотипа антитела. Примеры эффекторных функций антител включают: связывание C1q и комплемент-зависимую цитотоксичность (CDC); Связывание с Fc-рецептором; антителозависимую клеточно-опосредованную цитотоксичность (ADCC); фагоцитоз; отрицательную регуляцию рецепторов клеточной поверхности (например, B-клеточного рецептора); и активацию В-клеток.
[0388] В некоторых вариантах осуществления антитело, например фрагмент антитела, может содержать по меньшей мере часть константной области иммуноглобулина, такую как один или более доменов константной области. В некоторых вариантах осуществления константные области включают константную область легкой цепи и/или константную область 1 тяжелой цепи (CH1). В некоторых вариантах осуществления антитело включает домен CH2 и/или CH3, такой как область Fc. В некоторых вариантах осуществления область Fc представляет собой область Fc человеческого IgG, такого как IgG1 или IgG4.
[0389] Термин «область Fc» в настоящем описании используется для определения С-концевой области тяжелой цепи иммуноглобулина, которая содержит, по меньшей мере, часть константной области. Термин включает области Fc с нативной последовательностью и варианты областей Fc. В одном варианте осуществления область Fc тяжелой цепи человеческого IgG простирается от Cys226 или от Pro230 до карбоксильного конца тяжелой цепи. Однако C-концевой лизин (Lys447) области Fc может присутствовать или не присутствовать. Если не указано иное, нумерация аминокислотных остатков в области Fc или константной области осуществляется в соответствии с системой нумерации EU, также называемой индексом EU, как описано в Kabat et al., Sequences of Proteins of Immunological Interest, 5th Ed. Public Health Service, National Institutes of Health, Bethesda, MD, 1991.
[0390] Термины «полноразмерное антитело», «интактное антитело» и «цельное антитело» используются в настоящем документе взаимозаменяемо для обозначения антитела, имеющего структуру, по существу сходную со структурой нативного антитела, или имеющего тяжелые цепи, которые содержат область Fc, как определено в настоящем описании.
[0391] «Выделенное» антитело представляет собой антитело, которое отделено от компонентов его естественной среды. В некоторых вариантах осуществления антитело очищают до чистоты более чем 95% или 99%, что определяется, например, электрофоретически (например, SDS-PAGE, изоэлектрическим фокусированием (IEF), капиллярным электрофорезом) или хроматографически (например, ионнообменная хроматография или ВЭЖХ с обращенной фазой). Для обзора способов оценки чистоты антител см., например, Flatman et al., J. Chromatogr. B 848: 79-87 (2007).
1. Варианты и модификации
[0392] В некоторых вариантах осуществления антитела или их антигенсвязывающие фрагменты включают одну или более вариаций аминокислот, например замен, делеций, вставок и/или мутаций, по сравнению с последовательностью антитела, описанной в настоящем документе. Иллюстративные варианты включают варианты, предназначенные для улучшения аффинности связывания и/или других биологических свойств антитела. Варианты аминокислотной последовательности антитела могут быть получены путем введения соответствующих модификаций в нуклеотидную последовательность, кодирующую антитело, или путем пептидного синтеза. Такие модификации включают, например, делеции и/или вставки и/или замены остатков в аминокислотных последовательностях антитела. Любая комбинация делеции, вставки и замены может быть сделана для достижения конечной конструкции, при условии, что конечная конструкция обладает желаемыми характеристиками, например, связыванием антигена.
[0393] В некоторых вариантах осуществления антитела включают одну или более аминокислотных замен, например, по сравнению с последовательностью антител, описанной в настоящем документе, и/или по сравнению с последовательностью природного репертуара, например человеческого репертуара. Сайты, представляющие интерес для заместительного мутагенеза, включают CDR и FR. Аминокислотные замены могут быть введены в представляющее интерес антитело, и продукты будут подвергнуты скринингу на предмет желаемой активности, например, сохраненного/улучшенного связывания антигена, сниженной иммуногенности, улучшенного периода полужизни и/или улучшенной эффекторной функции, такой как способность стимулировать антителозависимую клеточную цитотоксичность (ADCC) или комплементзависимую цитотоксичность (CDC).
[0394] В некоторых вариантах осуществления один или более остатков в CDR исходного антитела (например, гуманизированного или человеческого антитела) замещены. В некоторых вариантах осуществления замена выполняется для возвращения последовательности или положения в последовательности к последовательности зародышевой линии, такой как последовательность антитела, присутствующая в зародышевой линии (например, человеческой зародышевой линии), например, для уменьшения вероятности иммуногенности, например, после введения человеку.
[0395] В некоторых вариантах осуществления изменения делаются в «горячих точках» CDR, остатках, кодируемых кодонами, которые подвергаются мутации с высокой частотой во время процесса соматического созревания (см., например, Chowdhury, Methods Mol. Biol. 207: 179-196 (2008)) и/или остатках, которые контактируют с антигеном, причем полученный вариант VH или VL тестируют на аффинность связывания. Созревание аффинности путем конструирования и повторного отбора из вторичных библиотек описано, например, в Hoogenboom et al. Methods in Molecular Biology 178: 1-37 (O'Brien et al., ed., Human Press, Totowa, NJ, (2001)). В некоторых вариантах осуществления аффинного созревания разнообразие вводится в вариабельные гены, выбранные для созревания любым из множества способов (например, ПЦР пониженной точности, перетасовка цепей или олигонуклеотид-направленный мутагенез). Затем можно создать вторичную библиотеку и провести ее скрининг для выявления любых вариантов антител с желаемой аффинностью. Другой способ введения разнообразия включает CDR-направленные подходы, в которых несколько остатков CDR (например, 4-6 остатков за раз) рандомизированы. Остатки CDR, участвующие в связывании антигена, могут быть конкретно идентифицированы, например, с использованием аланинсканирующего мутагенеза или моделирования. В частности, CDR-H3 и CDR-L3 часто являются мишенями.
[0396] В некоторых вариантах осуществления замены, вставки или делеции могут происходить в одной или более CDR, при условии, что такие изменения существенно не снижают способность антитела связываться с антигеном. Например, в CDR могут быть сделаны консервативные изменения (например, консервативные замены, как предусмотрено в настоящем документе), которые существенно не снижают аффинность связывания. Такие изменения могут, например, присутствовать вне антигенсвязывающих остатков в CDR. В некоторых вариантах вариантах осуществления вариантов последовательностей VH и VL, представленных выше, каждая CDR либо не изменена, либо содержит не более одной, двух или трех аминокислотных замен.
[0397] Вставки аминокислотной последовательности включают амино- и/или карбоксиконцевые слияния длиной от одного остатка до полипептидов, содержащих сто или более остатков, а также вставки внутри последовательности одного или более аминокислотных остатков. Примеры концевых вставок включают антитело с N-концевым метионильным остатком. Другие варианты вставок молекулы антитела включают слияние с N- или C-концом антитела с ферментом или полипептидом, который увеличивает период полужизни антитела в сыворотке.
[0398] В некоторых вариантах осуществления антитело или его антигенсвязывающий фрагмент изменяют для увеличения или уменьшения степени, до которой антитело гликозилируется, например, путем удаления или вставки одного или более сайтов гликозилирования путем изменения аминокислотной последовательности и/или путем модификации олигосахарида(олигосахаридов), присоединенных к сайтам гликозилирования, например, с использованием определенных клеточных линий.
[0399] Иллюстративные модификации, варианты и клеточные линии описаны, например, в патентных публикациях No. US 2003/0157108, US 2004/0093621, US 2003/0157108; WO 2000/61739; WO 2001/29246; US 2003/0115614; US 2002/0164328; US 2004/0093621; US 2004/0132140; US 2004/0110704; US 2004/0110282; US 2004/0109865; WO 2003/085119; WO 2003/084570; WO 2005/035586; WO 2005/035778; WO2005/053742; WO2002/031140; Okazaki et al. J. Mol. Biol. 336: 1239-1249 (2004); Yamane-Ohnuki et al. Biotech. Bioeng. 87: 614 (2004). Ripka et al. Arch. Biochem. Biophys. 249: 533-545 (1986); Патентная заявка США № US 2003/0157108 A1, Presta, L; и WO 2004/056312 A1, Yamane-Ohnuki et al. Biotech. Bioeng. 87: 614 (2004); Kanda Y. et al., Biotechnol. Bioeng., 94 (4): 680-688 (2006); и WO2003/085107); WO 2003/011878 (Jean-Mairet et al.); Патент США № 6602684 (Umana et al.); и US 2005/0123546 (Umana et al.); WO 1997/30087 (Patel et al.); WO 1998/58964 (Raju, S.); и WO 1999/22764 (Raju, S.).
[0400] Среди модифицированных антител имеются антитела, имеющие одну или более аминокислотных модификаций в области Fc, такие как антитела, имеющие последовательность области Fc человека или другую часть константной области (например, область Fc IgG1, IgG2, IgG3 или IgG4 человека), содержащую аминокислотную модификацию (например, замену) в одном или более положениях аминокислот.
[0401] Такие модификации могут быть сделаны, например, для улучшения периода полужизни, изменения связывания с одним или более типами Fc-рецепторов и/или изменения эффекторных функций.
[0402] Также среди вариантов присутствуют сконструированные антитела, содержащие цистеин, такие как «thioMAb», и другие сконструированные варианты по цистеину, в которых один или более остатков антитела замещены остатками цистеина для генерации реакционноспособных тиоловых групп в доступных сайтах, например, для использования в конъюгации агентов и линкер-агентов для получения иммуноконъюгатов. Сконструированные с использованием цистеина антитела описаны, например, в Патенты США № 7855275 и 7521541.
[0403] В некоторых вариантах осуществления антитела модифицированы, чтобы содержать дополнительные небелковые фрагменты, включая водорастворимые полимеры. Иллюстративные полимеры включают, но не ограничиваются ими, полиэтиленгликоль (ПЭГ), сополимеры этиленгликоль/пропиленгликоль, карбоксиметилцеллюлозу, декстран, поливиниловый спирт, поливинилпирролидон, поли-1,3-диоксолан, поли-1,3,6- триоксан, сополимер этилена/малеинового ангидрида, полиаминокислоты (гомополимеры или статистические сополимеры) и декстран или поли (н-винилпирролидон) полиэтиленгликоль, гомополимеры пропропиленгликоля, сополимеры пролипропиленоксида/этиленоксида, полиоксиэтилированные полиолы (например, глицерин), поливиниловый спирт и их смеси. Полиэтиленгликоль пропиональдегид может иметь преимущества при производстве благодаря его стабильности в воде. Полимер может иметь любую молекулярную массу и может быть разветвленным или неразветвленным. Количество полимеров, прикрепленных к антителу, может варьироваться, и если присоединено более одного полимера, они могут быть одинаковыми или разными молекулами. В общем, количество и/или тип полимеров, используемых для дериватизации, могут быть определены на основе соображений, включающих, но не ограничивающихся ими, конкретные свойства или функции антитела, подлежащего улучшению, независимо от того, будет ли производное антитела использоваться в терапии при определенные условия и т. д.
2. TCR-подобные CAR
[0404] В некоторых вариантах осуществления антитело или его антигенсвязывающая часть экспрессируются на клетках в виде части рекомбинантного рецептора, такого как рецептор антигена. Среди антигенных рецепторов присутствуют функциональные рецепторы, отличные от TCR-антигенных рецепторов, такие как химерные антигенные рецепторы (CAR). Как правило, CAR, содержащий антитело или антигенсвязывающий фрагмент, который проявляет TCR-подобную специфичность, направленную против пептида в контексте молекулы MHC, также может называться TCR-подобным CAR.
[0405] Таким образом, среди представленных связывающих молекул, например, HPV 16 E6 или E7-связывающих молекул, присутствуют антигенные рецепторы, такие как те, которые включают одно из представленных антител, например TCR-подобные антитела. В некоторых вариантах осуществления антигенные рецепторы и другие химерные рецепторы специфически связываются с областью или эпитопом E6 или E7 HPV16, такие как антигенные рецепторы, содержащие представленные антитела против E6 или E7 HPV 16 или фрагменты антител, например, TCR-подобные антитела. Среди антигенных рецепторов присутствуют функциональные рецепторы, отличные от TCR-антигенных рецепторов, такие как химерные антигенные рецепторы (CAR). Также представлены клетки, экспрессирующие CAR, и их применение в адоптивной клеточной терапии, такой как лечение заболеваний и расстройств, связанных с экспрессией E6 или E7 HPV 16.
[0406] Таким образом, в настоящем документе представлены TCR-подобные CAR, которые содержат молекулу, не относящуюся к TCR, которая проявляет специфичность Т-клеточных рецепторов, как например, для эпитопа Т-клеток или пептидного эпитопа, когда отображается или презентируется в контексте молекулы МНС. В некоторых вариантах осуществления TCR-подобный CAR может содержать антитело или его антигенсвязывающую часть, например TCR-подобное антитело, такое как описано в настоящем документе. В некоторых вариантах осуществления антитело или его антигенсвязывающая часть является реакционноспособной в отношении специфического пептидного эпитопа в контексте молекулы МНС, где антитело или фрагмент антитела могут дифференцировать конкретный пептид в контексте молекулы МНС от одной молекулы МНС, только конкретный пептид и, в некоторых случаях, нерелевантный пептид в контексте молекулы МНС. В некоторых вариантах осуществления антитело или его антигенсвязывающая часть могут проявлять более высокую аффинность связывания, чем Т-клеточный рецептор.
[0407] Иллюстративные антигенные рецепторы, включающие CAR, и способы конструирования и введения таких рецепторов в клетки включают те, которые описаны, например, в международных публикациях патентных заявок WO2000/14257, WO2013/126726, WO2012/129514, WO2014/031687, WO2013/166321, WO2013/071154, WO2013/123061, в публикациях патентных заявок США US2002/131960, US2013/287748, US2013/0149337, в Патентах США № 6451995, 7446190, 8252592, 8339645, 8398282, 7446179, 6410319, 7070995, 7265209, 7354762, 7446191, 8324353 и 8479118, а также в еврпейской патентной заявке EP 2537416 и/или описанные в Sadelain et al., Cancer Discov. 2013 April; 3(4): 388-398; Davila et al. (2013) PLOS ONE 8 (4): e61338; Turtle et al., Curr. Opin. Immunol., 2012 October; 24(5): 633-39; Wu et al., Cancer, 2012 March 18(2): 160-75. В некоторых аспектах антигенные рецепторы включают CAR, как описано в Патенте США № 7446190 и те, которые описаны в публикации международной заявки на патент: WO2014/055668 A1. Примеры CAR включают CAR, как описано в любой из вышеупомянутых публикаций, таких как WO2014/031687, US 8339645, US 7446179, US 2013/0149337, Патент США № 7446190, Патент США № 8389282, например, и в котором антигенсвязывающая часть, например scFv, заменена антителом, например, как предусмотрено в настоящем описании.
[0408] В некоторых вариантах осуществления CAR, как правило, включают внеклеточный антигенсвязывающий домен (или лиганд-связывающий), в том числе в виде антитела или его антигенсвязывающего фрагмента, специфичного для пептида в контексте молекулы МНС, связанный с одним или более внутриклеточными сигнальными компонентами, в некоторых аспектах через линкеры и/или трансмембранные домены. В некоторых вариантах осуществления такие молекулы обычно могут имитировать или аппроксимировать сигнал через природный антигенный рецептор, такого как TCR, и, необязательно, сигнал через такой рецептор в комбинации с костимуляторным рецептором.
[0409] В некоторых вариантах осуществления CAR обычно включает в свою внеклеточную часть одну или более антигенсвязывающих молекул, таких как один или более антигенсвязывающих фрагментов, доменов или частей, или один или более вариабельных доменов антител, и/или молекулы антител. В некоторых вариантах осуществления CAR включает антигенсвязывающую часть или части молекулы антитела, такие как одноцепочечный фрагмент антитела (scFv), полученный из вариабельной области тяжелой цепи (VH) и вариабельной области легкой цепи (VL) моноклонального антитела (mAb). В некоторых вариантах осуществления CAR содержит TCR-подобное антитело, такое как антитело или антигенсвязывающий фрагмент (например, scFv), которое специфически распознает пептидный эпитоп, презентированный на поверхности клетки в контексте молекулы MHC.
[0410] В некоторых аспектах компонент антигенспецифического связывания или распознавания связан с одним или более трансмембранными и внутриклеточными сигнальными доменами. В некоторых вариантах осуществления CAR включает трансмембранный домен, слитый с внеклеточным доменом CAR. В одном варианте осуществления используется трансмембранный домен, который естественным образом ассоциирован с одним из доменов в CAR. В некоторых случаях трансмембранный домен отбирают или модифицируют путем замены аминокислот, чтобы избежать связывания таких доменов с трансмембранными доменами одного и того же или разных поверхностных мембранных белков, чтобы минимизировать взаимодействия с другими членами рецепторного комплекса.
[0411] Трансмембранный домен в некоторых вариантах осуществления происходит из природного или синтетического источника. Если источник является природным, домен в некоторых аспектах происходит из любого мембраносвязанного или трансмембранного белка. Трансмембранные области включают области, происходящие из (т.е. которые содержат по меньшей мере трансмембранную область(области)) альфа-, бета- или дзета-цепи Т-клеточного рецептора, CD28, CD3-эпсилон, CD45, CD4, CD5, CD8, CD9, CD16, CD22, CD33, CD37, CD64, CD80, CD86, CD134, CD137, CD154. Альтернативно, трансмембранный домен в некоторых вариантах осуществления является синтетическим. В некоторых аспектах синтетический трансмембранный домен содержит преимущественно гидрофобные остатки, такие как лейцин и валин. В некоторых аспектах триплет фенилаланина, триптофана и валина будет присутствовать на каждом конце синтетического трансмембранного домена.
[0412] В некоторых вариантах осуществления присутствует короткий олиго- или полипептидный линкер, например, линкер длиной от 2 до 10 аминокислот, такой как линкер, содержащий глицины и серины, например глицин-сериновый дублет, и образует связь между трансмембранным доменом и цитоплазматическим сигнальным доменом CAR.
[0413] В некоторых вариантах осуществления CAR, например TCR-подобный CAR, такой как его часть антитела, дополнительно включает спейсер, который может представлять собой или включать по меньшей мере часть константной области иммуноглобулина или ее вариант или модифицированную версию, такую как шарнирная область, например шарнирная область IgG4, и/или область CH1/CL и/или Fc. В некоторых вариантах осуществления константная область или часть присходит из человеческого IgG, такого как IgG4 или IgG1. В некоторых аспектах часть константной области служит спейсерной областью между компонентом распознавания антигена, например scFv, и трансмембранным доменом. Спейсер может иметь длину, которая обеспечивает повышенную чувствительность клетки после связывания антигена по сравнению с отсутствием спейсера. В некоторых примерах спейсер имеет длину, составляющую или примерно составляющую 12 аминокислот или не более чем 12 аминокислот. Иллюстративные спейсеры включают те, которые содержат по меньшей мере примерно от 10 до 229 аминокислот, примерно от 10 до 200 аминокислот, примерно от 10 до 175 аминокислот, примерно от 10 до 150 аминокислот, примерно от 10 до 125 аминокислот, примерно от 10 до 100 аминокислот, примерно от 10 до 75 аминокислот, примерно от 10 до 50 аминокислот, примерно от 10 до 40 аминокислот, примерно от 10 до 30 аминокислот, примерно от 10 до 20 аминокислот или примерно от 10 до 15 аминокислот, и включая любое целое число между конечными точками любого из перечисленных диапазонов. В некоторых вариантах осуществления спейсерная область содержит примерно 12 аминокислот или менее, примерно 119 аминокислот или менее или примерно 229 аминокислот или менее. Иллюстративные спейсеры включают только шарнир IgG4, шарнир IgG4, связанный с доменами СН2 и СН3, или шарнир IgG4, связанный с доменом СН3. Иллюстративные спейсеры включают, но не ограничиваются ими, те, что описаны в Hudecek et al. (2013) Clin. Cancer Res., 19: 3153 или в публикации международной патентной заявки № WO2014/031687.
[0414] В некоторых вариантах осуществления константная область или часть присходит из человеческого IgG, такого как IgG4 или IgG1. В некоторых вариантах осуществления спейсер имеет последовательность ESKYGPPCPPCP (представленную в SEQ ID NO: 268) и кодируется последовательностью, представленной в SEQ ID NO: 269. В некоторых вариантах осуществления спейсер имеет последовательность, представленную в SEQ ID NO: 270. В некоторых вариантах осуществления спейсер имеет последовательность, представленную в SEQ ID NO: 271. В некоторых вариантах осуществления константная область или часть происходит из IgD. В некоторых вариантах осуществления спейсер имеет последовательность, представленную в SEQ ID NO: 272. В некоторых вариантах осуществления спейсер имеет последовательность аминокислот, которая имеет, по меньшей мере, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичности последовательности с любой из SEQ ID NO: 268, 270, 271 или 272.
[0415] Домен распознавания антигена обычно связан с одним или более внутриклеточными сигнальными компонентами, такими как сигнальные компоненты, которые имитируют активацию через комплекс антигенного рецептора, такой как комплекс TCR, в случае CAR, и/или сигнал через другой рецептор клеточной поверхности. Таким образом, в некоторых вариантах осуществления антитело или его антигенсвязывающий фрагмент связаны с одним или более трансмембранными и внутриклеточными сигнальными доменами. В некоторых вариантах осуществления трансмембранный домен слит с внеклеточным доменом. В одном варианте осуществления используется трансмембранный домен, который естественным образом ассоциирован с одним из доменов в рецепторе, например, CAR. В некоторых случаях трансмембранный домен отбирают или модифицируют путем замены аминокислот, чтобы избежать связывания таких доменов с трансмембранными доменами одного и того же или разных поверхностных мембранных белков, чтобы минимизировать взаимодействия с другими членами рецепторного комплекса.
[0416] Трансмембранный домен в некоторых вариантах осуществления происходит из природного или синтетического источника. Если источник является природным, домен в некоторых аспектах происходит из любого мембраносвязанного или трансмембранного белка. Трансмембранные области включают области, происходящие из (то есть включают по меньшей мере трансмембранную область (области)) альфа-, бета- или дзета-цепи Т-клеточного рецептора, CD28, CD3-эпсилон, CD45, CD4, CD5, CD8, CD9, CD 16, CD22, CD33, CD37, CD64, CD80, CD86, CD134, CD137, CD154. Альтернативно, трансмембранный домен в некоторых вариантах осуществления является синтетическим. В некоторых аспектах синтетический трансмембранный домен содержит преимущественно гидрофобные остатки, такие как лейцин и валин. В некоторых аспектах триплет фенилаланина, триптофана и валина будет присутствовать на каждом конце синтетического трансмембранного домена. В некоторых вариантах осуществления связь осуществляется линкерами, спейсерами и/или трансмембранным доменом (доменами).
[0417] К внутриклеточным сигнальным доменам относятся те, которые имитируют или аппроксимируют сигнал через рецептор природного антигена, сигнал через такой рецептор в комбинации с костимулирующим рецептором и/или сигнал только через один костимулирующий рецептор. В некоторых вариантах осуществления присутствует короткий олиго- или полипептидный линкер, например, линкер длиной от 2 до 10 аминокислот, такой как линкер, содержащий глицины и серины, например глицин-сериновый дублет, и образует связь между трансмембранным доменом и цитоплазматическим сигнальным доменом CAR.
[0418] CAR обычно включает, по меньшей мере, один внутриклеточный сигнальный компонент или компоненты. В некоторых вариантах осуществления CAR включает внутриклеточный компонент комплекса TCR, такой как цепь CD3+TCR, которая обеспечивает активацию Т-клеток и цитотоксичность, например, цепь CD3-дзета. Таким образом, в некоторых аспектах антигенсвязывающая молекула связана с одним или более клеточными сигнальными модулями. В некоторых вариантах осуществления клеточные сигнальные модули включают трансмембранный домен CD3, внутриклеточные сигнальные домены CD3 и/или трансмембранные домены других CD. В некоторых вариантах осуществления CAR дополнительно включает часть одной или более дополнительных молекул, таких как Fc-рецептор γ, CD8, CD4, CD25 или CD16. Например, в некоторых аспектах CAR включает химерную молекулу между CD3-дзета (CD3-ζ) или Fc-рецептором γ и CD8, CD4, CD25 или CD16.
[0419] В некоторых вариантах осуществления после лигирования CAR цитоплазматический домен или внутриклеточный сигнальный домен CAR активирует по меньшей мере одну из нормальных эффекторных функций или ответов иммунной клетки, например, T-клетки, сконструированные для экспрессии CAR. Например, в некоторых контекстах CAR индуцирует функцию T-клетки, такую как цитолитическая активность или Т-хелперная активность, как например, секреция цитокинов или других факторов. В некоторых вариантах осуществления вместо интактной иммуностимулирующей цепи используется, например, укороченная часть внутриклеточного сигнального домена антигенного рецепторного компонента или костимулирующей молекулы, если она передает сигнал эффекторной функции. В некоторых вариантах осуществления внутриклеточный сигнальный домен или домены включают цитоплазматические последовательности Т-клеточного рецептора (TCR), а в некоторых аспектах также последовательности корецепторов, которые в естественном контексте действуют совместно с таким рецептором, инициируя сигнальную трансдукцию рекрутирования антигенного рецептора, и/или любое производное или вариант таких молекул, и/или любую синтетическую последовательность, которая обладает такой же функциональной способностью.
[0420] В контексте природного TCR для полной активации обычно требуется не только передача сигналов через TCR, но и костимулирующий сигнал. Таким образом, в некоторых вариантах осуществления, чтобы способствовать полной активации, компонент для генерации вторичного или костимулирующего сигнала также включен в CAR. В других вариантах осуществления CAR не включает в себя компонент для генерации костимулирующего сигнала. В некоторых аспектах дополнительный CAR экспрессируется в той же клетке и обеспечивает компонент для генерации вторичного или костимулирующего сигнала. В некоторых аспектах клетка содержит первый CAR, который содержит сигнальные домены для индукции первичного сигнала, и второй CAR, который связывается со вторым антигеном и содержит компонент для генерации костимулирующего сигнала. Например, первый CAR может быть активирующим CAR, а второй CAR может быть костимулирующим CAR. В некоторых аспектах оба CAR должны быть лигированы, чтобы индуцировать конкретную эффекторную функцию в клетке, которая может обеспечить специфичность и селективность для типа клетки-мишени.
[0421] Т-клеточная активация в некоторых аспектах описывается как опосредуемая двумя классами цитоплазматических сигнальных последовательностей: теми, которые инициируют антиген-зависимую первичную активацию через TCR (первичные цитоплазматические сигнальные последовательности), и теми, которые действуют антиген-независимым образом, чтобы обеспечить вторичный или костимулирующий сигнал (вторичные цитоплазматические сигнальные последовательности). В некоторых аспектах CAR включает в себя один или оба из таких сигнальных компонентов.
[0422] В некоторых аспектах CAR включает первичную цитоплазматическую сигнальную последовательность, которая регулирует первичную активацию комплекса TCR. Первичные цитоплазматические сигнальные последовательности, которые действуют стимулирующим образом, могут содержать сигнальные мотивы, известные как иммунорецепторные мотивы активации на основе тирозина или ITAM. Примеры ITAM, содержащих первичные цитоплазматические сигнальные последовательности, включают последовательности, полученные из TCR или CD3-дзета, FcR-гамма, CD3-гамма, CD3-дельта или CD3-эпсилон. В некоторых вариантах осуществления цитоплазматическая сигнальная молекула(молекулы) в CAR содержит (содержат) цитоплазматический сигнальный домен, его часть или последовательность, происходящую из CD3-дзета.
[0423] В некоторых вариантах осуществления CAR включает в себя сигнальный домен и/или трансмембранную часть костимулирующего рецептора, такого как CD28, 4-1BB, OX40, DAP10 и ICOS. В некоторых аспектах один и тот же CAR включает как активирующий, так и костимулирующий компоненты; в других аспектах активирующий домен обеспечивается одним CAR, тогда как костимулирующий компонент обеспечивается другим CAR, распознающим другой антиген.
[0424] В некоторых вариантах осуществления активирующий домен включен в один CAR, тогда как костимулирующий компонент обеспечивается другим химерным рецептором, распознающим другой антиген. В некоторых вариантах осуществления CAR включают активирующие или стимулирующие CAR и костимулирующие рецепторы, оба экспрессируются на одной и той же клетке (см. WO2014/055668). В некоторых аспектах рецептор, содержащий антитело против Е6 или Е7 HPV 16, является стимулирующим или активирующим CAR; в других аспектах это костимулирующий рецептор. В некоторых вариантах осуществления клетки дополнительно включают ингибирующие CAR (iCAR, см. Fedorov et al., Sci. Transl. Medicine, 5 (215) (December, 2013)), такой как ингибирующий рецептор, распознающий пептидный эпитоп, отличный от E6 HPV 16 или E7 HPV 16, посредством чего активирующий сигнал, доставляемый через CAR, нацеленный на HPV 16, уменьшается или ингибируется связыванием ингибирующий CAR с его лигандом, например, для уменьшения эффектов ошибочного нацеливания.
[0425] В некоторых вариантах осуществления клетка, экспрессирующая представленный TCR или другую связывающую молекулу, дополнительно экспрессирует дополнительный рецептор, такой как рецептор, способный доставлять костимулирующий сигнал или сигнал, стимулирующий выживание, такой как костимулирующий рецептор (см. WO2014/055668), и/или для блокирования или изменения результата ингибирующего сигнала, такого как сигнал, обычно доставляемый посредством иммунной контрольной точки, или другую иммуноингибирующую молекулу, такую как экспрессируемая в микроокружении опухоли, например, для повышения эффективности таких сконструированных клеток. См., например, Tang et al., Am J Transl Res. 2015; 7 (3): 460-473. В некоторых вариантах осуществления клетка может дополнительно включать один или более других экзогенных или рекомбинантных или сконструированных компонентов, таких как один или более экзогенных факторов и/или костимулирующих лигандов, которые экспрессируются клетками или секретируются ими и могут стимулировать функцию, например, в микроокружении. Примеры таких лигандов и компонентов включают, например, TNFR и/или рецепторы или лиганды семейства Ig, например 41BBL, CD40, CD40L, CD80, CD86, цитокины, хемокины и/или антитела или другие молекулы, такие как scFv. См., например, публикацию патентной заявки № WO2008121420 A1, WO2014134165 A1, US20140219975 A1. В некоторых вариантах осуществления клетки содержат один или более ингибирующих рецепторов (iCAR, см. Fedorov et al., Sci. Transl. Medicine, 5 (215) (December, 2013)), таких как те, которые связываются с лигандом или антигеном, не ассоциированными с заболеванием или состоянием или не экспрессируемыми при таких заболеваниях или состояниях.
[0426] В некоторых вариантах осуществления внутриклеточный сигнальный домен содержит трансмембранный домен CD28 и сигнальный домен, связанный с внутриклеточным доменом CD3 (например, CD3-дзета). В некоторых вариантах осуществления внутриклеточный сигнальный домен содержит химерные костимулирующие домены CD28 и CD137 (4-1BB, TNFRSF9), связанные с внутриклеточным доменом CD3-дзета.
[0427] В некоторых вариантах осуществления CAR включает один или более, например, два или более костимулирующих домена и домен активации, например, первичный домен активации, в цитоплазматической части. Иллюстративные CAR включают внутриклеточные компоненты CD3-дзета, CD28 и 4-1BB.
[0428] В некоторых вариантах осуществления клетка, экспрессирующая CAR или другой антигенный рецептор, дополнительно включает маркер, такой как маркер клеточной поверхности, который может использоваться для подтверждения трансдукции или конструирования клетки для экспрессии рецептора, такого как укороченная версия рецептора клеточной поверхности, такого как укороченный EGFR (tEGFR). Иллюстративные суррогатные маркеры могут включать укороченные формы полипептидов клеточной поверхности, такие как укороченные формы, которые не функциональны и не трансдуцируют или не способны к трансдукции сигнала, или сигнала, обычно трансдуцируемого полноразмерной формой полипептида клеточной поверхности, и/или не интернализуются или не способны интернализоваться. Иллюстративные укороченные полипептиды клеточной поверхности, включая укороченные формы факторов роста или других рецепторов, такие как укороченный рецептор 2 эпидермального фактора роста человека (tHER2), укороченный рецептор эпидермального фактора роста (tEGFR, иллюстративная последовательность tEGFR, представленная в SEQ ID NO: 273 или 343) или простат-специфический мембранный антиген (PSMA) или его модифицированная форма. tEGFR может содержать эпитоп, распознаваемый антителом цетуксимаб (Erbitux®) или другим терапевтическим антителом против EGFR или связывающей молекулой, которые можно использовать для идентификации или отбора клеток, которые были сконструированы с помощью конструкции tEGFR и кодируемого экзогенного белка, и/или для устранения или отделения клеток, экспрессирующих кодируемый экзогенный белок. См. Патент США № 8802374 и Liu et al., Nature Biotech. 2016 April; 34 (4): 430-434). В некоторых аспектах маркер, например суррогатный маркер включает весь или часть (например, укороченную форму) CD34, NGFR, CD19 или укороченный CD19, например укороченный CD19, не являющийся человеческим, или рецептор эпидермального фактора роста (например, tEGFR). В некоторых вариантах осуществления маркер представляет собой или содержит флуоресцентный белок, такой как зеленый флуоресцентный белок (GFP), усиленный зеленый флуоресцентный белок (EGFP), такой как супер-кратный GFP (sfGFP), красный флуоресцентный белок (RFP), такой как tdTomato, mCherry, mStrawberry, AsRed2, DsRed или DsRed2, голубой флуоресцентный белок (CFP), сине-зеленый флуоресцентный белок (BFP), усиленный синий флуоресцентный белок (EBFP) и желтый флуоресцентный белок (YFP) и их варианты, включая видовые варианты, мономерные варианты и кодон-оптимизированные и/или улучшенные варианты флуоресцентных белков. В некоторых вариантах осуществления маркер представляет собой или содержит фермент, такой как люцифераза, ген lacZ из E. coli, щелочную фосфатазу, секретируемую эмбриональную щелочную фосфатазу (SEAP), хлорамфениколацетилтрансферазу (CAT). Иллюстративные светоизлучающие репортерные гены включают люциферазу (luc), β-галактозидазу, хлорамфениколацетилтрансферазу (CAT), β-глюкуронидазу (GUS) или их варианты.
[0429] В некоторых вариантах осуществления маркер является маркером селекции. В некоторых вариантах осуществления маркер селекции представляет собой или содержит полипептид, который придает устойчивость к экзогенным агентам или лекарственным средствам. В некоторых вариантах осуществления маркер селекции представляет собой ген устойчивости к антибиотику. В некоторых вариантах осуществления маркер селекции представляет собой ген устойчивости к антибиотику, придающий устойчивость к антибиотику клетке млекопитающего. В некоторых вариантах осуществления маркер селекции представляет собой или содержит ген устойчивости к пуромицину, ген устойчивости к гигромицину, ген устойчивости к бластидину, ген устойчивости к неомицину, ген устойчивости к генетицину или ген устойчивости к зеоцину или их модифицированную форму.
[0430] В некоторых аспектах маркер включает весь или часть (например, укороченную форму) CD34, NGFR или рецептора эпидермального фактора роста (например, tEGFR). В некоторых вариантах осуществления нуклеиновая кислота, кодирующая маркер, функционально связана с полинуклеотидом, кодирующим последовательность линкера, такую как расщепляемая последовательность линкера, например, T2A. Смотри WO2014031687. [0480] В некоторых вариантах осуществления введение конструкции, кодирующей CAR и EGFRt, разделенных переключателем рибосомы T2A, может экспрессировать два белка из одной и той же конструкции, так что EGFRt можно использовать в качестве маркера для детектирования клеток, экспрессирующих такую конструкцию. В некоторых вариантах осуществления маркер и, необязательно, линкерная последовательность могут быть любыми, как описано в опубликованной патентной заявке №. WO2014031687. Например, маркер может быть укороченным EGFR (tEGFR), который, необязательно, связан с последовательностью линкера, такой как расщепляемая последовательность линкера T2A. Иллюстративный полипептид для укороченного EGFR (например, tEGFR) содержит последовательность аминокислот, представленную в SEQ ID NO: 273 или 343, или последовательность аминокислот, которая имеет по меньшей мере 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичности последовательности с SEQ ID NO: 273 или 343. Иллюстративная линкерная последовательность T2A содержит последовательность аминокислот, представленную в SEQ ID NO: 211 или 274, или последовательность аминокислот, которая имеет по меньшей мере 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичности последовательности с SEQ ID NO: 211 или 274.
[0431] В некоторых вариантах осуществления маркер представляет собой молекулу, например белок клеточной поверхности или его часть, которые не обнаружены в природе в Т-клетках или не обнаружены в природе на поверхности Т-клеток.
[0432] В некоторых вариантах осуществления молекула представляет собой не собственную молекулу, например, не-собственный белок, то есть такие, которые не распознаются как «свои» иммунной системой хозяина, в которую будут адоптивно переноситься клетки.
[0433] В некоторых вариантах осуществления маркер не выполняет терапевтической функции и/или не производит никакого эффекта, кроме как для использования в качестве маркера для генетического конструирования, например, для отбора успешно сконструированных клеток. В других вариантах осуществления маркер может представлять собой терапевтическую молекулу или молекулу, иным образом оказывающую некоторый желаемый эффект, как например лиганд для клетки, встречающийся in vivo, такой как молекула костимулирующей или иммунной контрольной точки, для усиления и/или ослабления ответов клеток при адоптивном переносе и встрече с лигандом.
[0434] В некоторых случаях CAR относятся к CAR первого, второго и/или третьего поколения. В некоторых аспектах CAR первого поколения представляет собой тот, который обеспечивает только сигнал, индуцированный CD3-цепью, при связывании антигена; в некоторых аспектах CAR второго поколения представляют собой такие, которые обеспечивают такой сигнал и костимулирующий сигнал, например, такой, который включает внутриклеточный сигнальный домен из костимулирующего рецептора, такого как CD28 или CD137; в некоторых аспектах CAR третьего поколения в некоторых аспектах представляет собой тот, который включает множество костимулирующих доменов различных костимулирующих рецепторов.
[0435] В некоторых вариантах осуществления химерный антигенный рецептор включает внеклеточную часть, содержащую TCR-подобное антитело или фрагмент, описанный в настоящем документе, и внутриклеточный сигнальный домен. В некоторых вариантах осуществления антитело или его фрагмент включают scFv, а внутриклеточный домен содержит ITAM. В некоторых аспектах внутриклеточный сигнальный домен включает в себя сигнальный домен дзета-цепи CD3-дзета (CD3ζ). В некоторых вариантах осуществления химерный антигенный рецептор включает трансмембранный домен, связывающий внеклеточный домен и внутриклеточный сигнальный домен. В некоторых аспектах трансмембранный домен содержит трансмембранную часть CD28. Внеклеточный домен и трансмембранный могут быть связаны прямо или косвенно. В некоторых вариантах осуществления внеклеточный домен и трансмембранный связаны спейсером, таким как любой описанный в настоящем документе. В некоторых вариантах осуществления химерный антигенный рецептор содержит внутриклеточный домен костимулирующей молекулы Т-клеток, как например между трансмембранным доменом и внутриклеточным сигнальным доменом. В некоторых аспектах костимулирующей молекулой Т-клеток является CD28 или 41BB.
[0436] Например, в некоторых вариантах осуществления CAR содержит TCR-подобное антитело, например фрагмент антитела, как предусмотрено в настоящем документе, трансмембранный домен, который является или содержит трансмембранную часть CD28, или его функциональный вариант, и внутриклеточный сигнальный домен, содержащий сигнальная часть CD28, или его функциональный вариант, и сигнальная часть CD3 дзета или ее функциональный вариант. В некоторых вариантах осуществления CAR содержит TCR-подобное антитело, например фрагмент антитела, как предусмотрено в настоящем документе, трансмембранный домен, который является или содержит трансмембранную часть CD28, или его функциональный вариант, и внутриклеточный сигнальный домен, содержащий сигнальную часть 4-1BB, или его функциональный вариант, и сигнальная часть CD3-дзета или ее функциональный вариант. В некоторых таких вариантах осуществления CAR дополнительно включает спейсер, содержащий часть молекулы Ig, такой как молекула Ig человека, такую часть как шарнир Ig, например, шарнир IgG4, как например спейсер в виде шарнира.
[0437] В некоторых вариантах осуществления трансмембранный домен рецептора, например TCR-подобный CAR, представляет собой трансмембранный домен человеческого CD28 (например, регистр. No P01747.1) или его вариант, такой как трансмембранный домен, который содержит последовательность аминокислот, представленную в SEQ ID NO: 275, или последовательность аминокислот, которая имеет по меньшей мере 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичности последовательности с SEQ ID NO: 275. В некоторых вариантах осуществления часть CAR, содержащая трансмембранный домен, содержит последовательность аминокислот, представленную в SEQ ID NO: 276, или последовательность аминокислот, имеющую, по меньшей мере, или примерно 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичности последовательности с SEQ ID NO: 276.
[0438] В некоторых вариантах осуществления внутриклеточный сигнальный компонент(компоненты) CAR, например, TCR-подобный CAR, содержит внутриклеточный костимулирующий сигнальный домен CD28 человека или его функциональный вариант, или часть, такую как домен с заменой LL на GG в положениях 186-187 нативного белка CD28. Например, внутриклеточный сигнальный домен может содержать последовательность аминокислот, представленную в SEQ ID NO: 277 или 278, или последовательность аминокислот, которая имеет по меньшей мере 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичности последовательности с SEQ ID NO: 277 или 278. В некоторых вариантах осуществления внутриклеточный домен содержит внутриклеточный костимулирующий сигнальный домен 4-1BB (например, регистр. No Q07011.1) или его функциональный вариант или часть, такую как последовательность аминокислот, представленная в SEQ ID NO: 279, или последовательность аминокислот, которая имеет по меньшей мере 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичности последовательности с SEQ ID NO: 279.
[0439] В некоторых вариантах осуществления внутриклеточный сигнальный домен CAR, например, TCR-подобный CAR содержит стимулирующий сигнальный домен CD3-дзета человека или его функциональный вариант, как например цитоплазматический домен 112 AA изоформы 3 человеческого CD3ζ (регистр. No: P20963.2) или сигнальный домен CD3-дзета, как описано в Патенте США № 7446190 или в Патенте США № 8911993. Например, в некоторых вариантах осуществления внутриклеточный сигнальный домен содержит последовательность аминокислот SEQ ID NO: 280, 281 или 282 или последовательность аминокислот, которая имеет по меньшей мере 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичности последовательности с SEQ ID NO: 280, 281 или 282.
[0440] В некоторых аспектах спейсер содержит только шарнирную область IgG, такую как только шарнир IgG4 или IgG1, такой как спейсер в виде только шарнира, представленный в SEQ ID NO: 268. В других вариантах осуществления спейсер представляет собой или содержит шарнир Ig, например шарнир, полученный из IgG4, необязательно связанный с доменами СН2 и/или СН3. В некоторых вариантах осуществления спейсер представляет собой шарнир Ig, например шарнир IgG4, связанный с доменами СН2 и СН3, как представлено в SEQ ID NO: 271. В некоторых вариантах осуществления спейсер представляет собой шарнир Ig, например шарнир IgG4, связанный только с доменом СН3, как представлено в SEQ ID NO: 270. В некоторых вариантах осуществления спейсер представляет собой или содержит последовательность, богатую глицином-серином, или другой гибкий линкер, такой как известные гибкие линкеры.
[0441] Например, в некоторых вариантах осуществления TCR-подобный CAR включает в себя TCR-подобное антитело или фрагмент, такие как любые из предложенных в настоящем описании, включая scFv, спейсер, такой как любой из спейсеров, содержащих Ig-петли, трансмембранный домен CD28, внутриклеточный сигнальный домен CD28 и сигнальный домен CD3 дзета. В некоторых вариантах осуществления TCR-подобный CAR включает в себя TCR-подобное антитело или фрагмент, такие как любые из предложенных в настоящем описании, включая scFv, спейсер, такой как любой из спейсеров, содержащих Ig-шарнир, трансмембранный домен CD28, внутриклеточный сигнальный домен CD28 и сигнальный домен CD3-дзета. В некоторых вариантах осуществления такие TCR-подобные конструкции CAR дополнительно включают в себя элемент пропуска рибосомы T2A и/или последовательность tEGFR, например, в 3'-области от CAR.
[0442] В некоторых вариантах осуществления такие конструкции CAR дополнительно включают в себя элемент пропуска рибосомы T2A и/или последовательность tEGFR, например, в 3'-области от CAR, как представлено в SEQ ID NO: 211 или 274, и последовательность tEGFR, представленную в SEQ ID NO: 273 или 343, или последовательность аминокислот, которая имеет по меньшей мере 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичности последовательности с SEQ ID NO: 211, 273, 343 или 274.
[0443] В некоторых вариантах осуществления CAR включает антитело или фрагмент E6 или E7 HPV 16, такие как любое из антител пртив E6 или E7 HPV16, включая sdAb (например, содержащие только область VH) и scFv, описанные в настоящем документе, спейсер, такой как любой из спейсеров, содержащих Ig-шарнир, трансмембранный домен CD28, внутриклеточный сигнальный домен CD28 и сигнальный домен CD3-дзета. В некоторых вариантах осуществления CAR включает антитело или фрагмент HPV 16, такие как любое из антител против E6 или E7 HPV 16, включая sdAb и scFv, описанные в настоящем документе, спейсер, такой как любой из спейсеров, содержащих Ig-шарнир, трансмембранный домен CD28, внутриклеточный сигнальный домен CD28 и сигнальный домен CD3-дзета. В некоторых вариантах осуществления такие конструкции CAR дополнительно включают в себя элемент пропуска рибосомы T2A и/или последовательность tEGFR, например, в 3'-области от CAR.
3. Иллюстративные особенности связывающих молекул и сконструированных клеток
[0444] В некоторых аспектах предусмотрены связывающие молекулы, например, TCR или TCR-подобные CAR имеют одну или более указанных функциональных особенностей, таких как свойства связывания, включая связывание с конкретными эпитопами, отсутствие нецелевого связывания или активности и/или специфические аффинности связывания. В некоторых вариантах осуществления любой один или более признаков представленного TCR можно оценить путем экспрессии TCR, например, путем введения одной или более нуклеиновых кислот, кодирующих TCR, в T-клетку, такую как первичная T-клетка или T-клеточная линия. В некоторых вариантах осуществления Т-клеточная линия представляет собой клетку Jurkat или клеточную линию, полученную из Jurkat. Примером клеточной линии, полученной из Jurkat, является клеточная линия J.RT3-T3.5 (ATCC® TIB-153™), полученная путем обработки клеточной линии лейкоза Jurkat с помощью радиационного мутагенеза и отрицательного отбора с помощью моноклонального антитела против OKT3 (см. Weiss). & Stobo, J. Ex. Med. 160 (5): 1284-1299 (1984)).
[0445] В некоторых вариантах осуществления предложенные связывающие молекулы способны связываться с пептидным эпитопом HPV16, например, эпитопом HPV 16, Е6 или Е7, такой как описанный выше, по меньшей мере с определенной аффинностью, измеренной любым из ряда известных способов. В некоторых вариантах осуществления пептидный эпитоп представляет собой пептид в контексте молекулы МНС или лиганда. В некоторых вариантах осуществления аффинность представлена константой равновесия диссоциации (KD) или константой ассоциации (ka). В некоторых вариантах осуществления аффинность представлена EC50.
[0446] В некоторых вариантах осуществления связывающая молекула, например TCR, связывается, например, специфически связывается, с пептидным эпитопом, например, в комплексе с молекулой MHC, с аффинностью или KA (то есть константой равновесия ассоциации конкретного связывающего взаимодействия с единицами 1/M, равной отношению скорости ассоциации [kon или ka] к скорости диссоциации [koff или kd] для этой реакции ассоциации, предполагающей бимолекулярное взаимодействие), равной или превышающей 105 М-1. В некоторых вариантах осуществления TCR или его фрагмент проявляют аффинность связывания пептидного эпитопа с KD (то есть с константой равновесия диссоциации конкретного связывающего взаимодействия с единицами M; равной отношению скорости диссоциации [koff или kd] к скорости ассоциации [kon или ka] для этой реакции ассоциации, при условии бимолекулярного взаимодействия), равной или меньшей 10-5 М. Например, константа равновесия диссоциации KD находится в диапазоне от или примерно от 10-5 М до или примерно до 10-12 М, например, от или примерно от 10-6 М до или примерно до 10-10 М, от или примерно от 10-7 М до или прмерно до 10-11 М, от или примерно от 10-6 М до или примерно до 10-8 М, или от или примерно от 10-7 М до или примерно до 10-8 М. Скорость ассоциации (константа скорости ассоциации; kon или ka; единицы 1/Мс) и скорость диссоциации (константа скорости диссоциации; koff или kd; единицы 1/с) может быть определена с использованием любого из методов анализа, известных в данной области, например, поверхностного плазмонного резонанса (SPR).
[0447] В некоторых вариантах осуществления аффинность связывания может быть классифицирована как высокая аффинность или низкая аффинность. В некоторых случаях связывающая молекула (например, TCR), которая проявляет связывание от низкой до умеренной аффинности, демонстрирует KA до 107 М-1, до 106 М-1, до 105 М-1. В некоторых случаях связывающая молекула (например, TCR), которая проявляет высокую аффинность связывания с конкретным эпитопом, взаимодействует с таким эпитопом с помощью KA, составляющей по меньшей мере 107 М-1, по меньшей мере 108 М-1, по меньшей мере 109 М-1, по меньшей мере 1010 М-1, по меньшей мере 1011 М-1, по меньшей мере 1012 М-1 или по меньшей мере 1013 М-1. В некоторых вариантах осуществления аффинность связывания (EC50) и/или константа диссоциации молекулы, связывающей пептидный эпитоп HPV 16, E6 или E7, составляет от или примерно от 0,1 нМ до 1 мкМ, от 1 нМ до 1 мкМ, от 1 нМ до 500 нМ, от 1 нМ до 100 нМ, от 1 нМ до 50 нМ, от 1 нМ до 10 нМ, от 10 нМ до 500 нМ, от 10 нМ до 100 нМ, от 10 нМ до 50 нМ, от 50 нМ до 500 нМ, от 50 нМ до 100 нМ или от 100 до 500 нМ. В некоторых вариантах осуществления аффинность связывания (EC50) и/или константа диссоциации молекулы, связывающей пептидный эпитоп HPV 16, E6 или E7, равна или примерно равна или составляет менее чем или примерно 1 мкМ, 500 нм, 100 нМ, 50 нМ, 40 нМ, 30 нМ, 25 нМ, 20 нМ, 19 нМ, 18 нМ, 17 нМ, 16 нМ, 15 нМ, 14 нМ, 13 нМ, 12 нМ, 11 нМ, 10 нМ, 9 нМ, 8 нМ, 7 нМ, 6 нМ, 5 нМ, 4 нМ, 3 нМ, 2 нМ или 1 нМ.
[0448] Известно множество анализов для оценки аффинности связывания и/или определения того, связывается ли связывающая молекула со специфическим лигандом (например, пептидом в контексте молекулы МНС). Специалист в данной области может определить аффинность связывания связывающей молекулы, например TCR, для эпитопа Т-клеток полипептида-мишени, например, с помощью любого из ряда анализов связывания, которые хорошо известны в данной области. Например, в некоторых вариантах осуществления аппарат BIAcore может использоваться для определения константы связывания комплекса между двумя белками. Константа диссоциации для комплекса может быть определена путем мониторинга изменений показателя преломления во времени при прохождении буфера по чипу. Другие подходящие анализы для измерения связывания одного белка с другим включают, например, иммуноанализы, такие как иммуноферментные анализы (ИФА) и радиоиммуноанализы (RIA), или определение связывания путем мониторинга изменения спектроскопических или оптических свойств белков. через флуоресценцию, УФ-поглощение, круговой дихроизм или ядерный магнитный резонанс (ЯМР). Другие иллюстративные анализы включают, но не ограничиваются ими, вестерн-блот, ИФА, аналитическое ультрацентрифугирование, спектроскопию и анализ поверхностного плазмонного резонанса (Biacore®) (см., Например, Scatchard et al., Ann. N.Y. Acad. Sci. 51:660, 1949; Wilson, Science 295:2103, 2002; Wolff et al., Cancer Res. 53: 2560, 1993; и Патенты США № 5283173, 5468614 или эквивалент), проточную цитометрию, секвенирование и другие методы детектирования экспрессированных нуклеиновых кислот. В одном примере кажущуюся аффинность к TCR измеряют путем оценки связывания с различными концентрациями тетрамеров, например, с помощью проточной цитометрии с использованием меченых тетрамеров. В одном примере кажущуюся KD TCR измеряют с использованием 2-кратных разведений меченых тетрамеров в диапазоне концентраций с последующим определением кривых связывания с помощью нелинейной регрессии, причем кажущуюся KD определяют как концентрацию лиганда, при которой получают половину максимального связывания.
[0449] В некоторых вариантах осуществления связывающие молекулы проявляют предпочтение связывания для распознавания антигеном HPV 16, E6- или E7-экспрессирующих клеток, по сравнению с HPV 16, E6- или E7-негативными клетками, такими как конкретные клетки, известные и/или описанные в настоящем документе для экспрессии HPV 16 E6 или E7, и которые, как известно, не экспрессируют E6 или E7 HPV 16. В некоторых вариантах осуществления предпочтение связывания наблюдается, когда измеряется значительно более высокая степень связывания с HPV 16 E6- или E7-экспрессирующими клетками по сравнению с клетками, экспрессирующими Е6 или Е7, не относящиеся к HPV 16. В некоторых вариантах осуществления детектируется кратное изменение степени связывания, например, измеренное по средней интенсивности флуоресценции в анализе на основе проточной цитометрии и/или константе диссоциации или EC50, для клеток, экспрессирующих E6 HPV 16 или E7 HPV 16, по сравнению с клетками, экспрессирующими Е6 или Е7, не относящиеся к HPV 16, составляет, по меньшей мере, 1,5 или 2, 3, 4, 5, 6 или более.
[0450] В некоторых вариантах осуществления связывающая молекула, например TCR не проявляет перекрестно-реактивного или нецелевого связывания, такого как нежелательное нецелевое связывание, например, нецелевое связывание с антигенами, присутствующими в здоровых или нормальных тканях или клетках. В некоторых вариантах осуществления связывающая молекула, например TCR распознает, например, специфически связывается только с одним пептидным эпитопом или антигенным комплексом, как например распознает только конкретный эпитоп HPV 16 Е6 или Е7, представленный в любой из SEQ ID NO: 232-239, или его антигенный комплекс. Таким образом, в некоторых вариантах осуществления представленные связывающие молекулы, например, TCR имеют пониженный риск вызывать нежелательные побочные эффекты, например, из-за распознавания нецелевого пептидного эпитопа.
[0451] В некоторых вариантах осуществления связывающая молекула, например TCR, не распознает, например, не связывает специфически, связанный с последовательностью пептидный эпитоп эпитопа E6 или E7 HPV 16, представленный в любой из SEQ ID NO: 232-239, т. е. не распознает эпитоп, имеющий некоторые общие аминокислоты с эпитопом HPV 16, E6 или E7, представленным в любой из SEQ ID NO: 232-239, как например, не распознает эпитоп, который отличается на 1, 2, 3, 4, 5 или 6 аминокислотных остатков из такого эпитопа, когда эпитопы выровнены. В некоторых вариантах осуществления связывающая молекула, например TCR, не распознает эпитоп, не относящийся к последовательности эпитопа E6 или E7 HPV 16, представленного в любой из SEQ ID NO: 232-239, то есть не распознает эпитоп, который существенно отличается по последовательности по сравнению с эпитопом HPC 16 E6 или E7, представленным в любой из SEQ ID NO: 232-239, как например, отличается более чем на 6, 7, 8, 9, 10 или более аминокислотных остатков от такого эпитопа при выравнивании эпитопов. В некоторых вариантах осуществления связывающая молекула, например TCR, не распознает эпитоп Е6 или Е7 HPV 16, представленный в любой из SEQ ID NO: 232-239, в контексте другого аллеля МНС, как например в контексте аллеля МНС, отличного от HLA-A2.
[0452] Как правило, специфическое связывание связывающей молекулы, например, TCR с пептидным эпитопом, например в комплексе с МНС определяется наличием антигенсвязывающего сайта, содержащего одну или более областей, определяющих комплементарность (CDR). В целом понятно, что специфическое связывание не означает, что конкретный пептидный эпитоп, например в комплексе с МНС является единственной вещью, с которой может связываться молекула МНС-пептид, поскольку также могут происходить неспецифические связывающие взаимодействия с другими молекулами. В некоторых вариантах осуществления связывание связывающей молекулы с пептидом в контексте молекулы МНС имеет более высокую аффинность, чем связывание с другими молекулами, например, другим пептидом в контексте молекулы МНС или нерелевантным (контрольным) пептидом в контексте молекулы МНС, как например, по меньшей мере, примерно в 2 раза, по меньшей мере, примерно в 10 раз, по меньшей мере, примерно в 20 раз, по меньшей мере, примерно в 50 раз или, по меньшей мере, примерно в 100 раз выше, чем аффинность связывания с другими молекулами.
[0453] В некоторых вариантах осуществления связывающую молекулу, например TCR, можно оценивать на безопасность или активность нецелевого связывания с использованием любого из ряда анализов скрининга, известных в данной области. В некоторых вариантах осуществления генерирование иммунного ответа на конкретную связывающую молекулу, например TCR, можно измерить в присутствии клеток, о которых известно, что они не экспрессируют пептидный эпитоп-мишень, таких как клетки, происходящие из нормальной ткани (тканей), аллогенной клеточные линии, которые экспрессируют один или более различных типов МНС, или ткани или клетки из других источников. В некоторых вариантах осуществления клетки или ткани включают нормальные клетки или ткани. Например, в некоторых случаях клетки или ткани могут включать мозг, мышцы, печень, толстую кишку, почку, легкое, яичник, плаценту, сердце, поджелудочную железу, простату, эпителий или кожу, яичко, надпочечник, кишечник, костный мозг или селезенку. В некоторых вариантах осуществления связывание с клетками можно тестировать в 2-мерных культурах. В некоторых вариантах осуществления связывание с клетками можно тестировать в 3-мерных культурах. В некоторых вариантах осуществления в качестве контроля ткани или клетки могут быть такими, которые, как известно, экспрессируют эпитоп-мишень. Иммунный ответ можно оценивать прямо или косвенно, например, путем оценки активации иммунных клеток, таких как Т-клетки (например, цитотоксической активности), продукции цитокинов (например, гамма-интерферона) или активации сигнального каскада.
[0454] В некоторых вариантах осуществления потенциальные нецелевые мишени могут быть идентифицированы путем выполнения гомологического сканирования генома человека с использованием конкретного эпитопа-мишени, например, для идентификации потенциальных эпитопов, связанных с последовательностью. В некоторых случаях база данных последовательностей белков может быть проанализирована для идентификации пептидов, сходных с пептидным эпитопом-мишенью. В некоторых вариантах осуществления, чтобы облегчить идентификацию потенциальных связанных с последовательностями эпитопов, представляющих интерес, сначала может быть идентифицирован мотив связывания. В некоторых вариантах осуществления мотив связывания может быть идентифицирован путем пептидного сканирования, такого как аланинсканирующий мутагенез эпитопа-мишени (например, эпитопа E6 HPV 16 или E7 HPV 16, представленного в любой из SEQ ID NO: 232-239), чтобы идентифицировать мотив связывания, распознаваемый связывающей молекулой, см., например, WO2014/096803. В некоторых вариантах осуществления мотив связывания может быть идентифицирован мутагенезом пептида-мишени, так что генерируется ряд мутантов, в которых каждая аминокислота или ее подмножество заменены на другой аминокислотный остаток, протестированный на его активность относительно исходного эпитопа-мишени, и идентифицируют те остатки, которые вовлечены или необходимы для связывания. В некоторых вариантах осуществления может быть получен ряд мутантов, в которых аминокислотный остаток в каждом положении эпитопа-мишени мутирован во все альтернативные аминокислоты. В некоторых случаях после идентификации мотива связывания (то есть аминокислотных остатков, которые не являются толерантными и участвуют в связывании или требуются для него), в базах данных белков можно искать белки, которые содержат мотив связывания.
[0455] В некоторых вариантах осуществления подходящие белковые базы данных включают, но не ограничиваются ими, UniProtKB/Swiss-Prot (http://www.uniprot.org/), Информационный ресурс по белкам (PI R) (http://pir.georgetown.edu/pirwww). /index.shtml) и/или Эталонная последовательность (RefSeq) (www.ncbi.nlm.nih.gov/RefSeq). Поиск пептидного мотива может быть выполнен с использованием любого из множества инструментов, которые можно найти на сайтах ресурсов по биоинформатике, таких как ExPASY (http://www.expasy.org/). Например, инструмент поиска ScanProsite идентифицирует определенные пользователем мотивы во всех белковых последовательностях в базе данных UniProtKB/Swiss-Prot Protein (De Castro et al. Nucleic Acids Res. 2006 Jul 1; 34 (Web Server issue):W362-5). В некоторых случаях поиск может проводиться для пептидов человеческого происхождения или организмов, которые обычно присутствуют у людей, таких как вирусные или бактериальные патогены или комменсальные бактерии.
[0456] В некоторых вариантах осуществления, если потенциальный нецелевой эпитоп идентифицирован, связывающая молекула, например, TCR, может быть изменена таким образом, чтобы больше не было перекрестной реактивности с нецелевым пептидом (пептид), сохраняя при этом связывание, предпочтительно с высокой аффинностью, с целевым пептидным эпитопом. Например, Т-клеточные рецепторы могут быть перепроектированы путем мутагенеза с использованием способов, описанных в WO 03/020763.
[0457] В некоторых вариантах осуществления связывающие молекулы, например сконструированные клетки, содержащие связывающие молекулы, например TCR, вызывают иммунный ответ на HPV 16. В некоторых вариантах осуществления цитотоксические T-лимфоциты (CTL) могут активироваться, когда клетки, содержащие связывающие молекулы, например TCR, контактируют с клетками-мишенями, такими как клетки, которые экспрессируют HPV 16, такие как E6 HPV 16 или E7 HPV 16. Например, клетки, содержащие TCR, могут индуцировать лизис клеток-мишеней, таких как HPV 16-экспрессирующие, например, HPV 16 E6- или E7-экспрессирующие клетки. В некоторых аспектах способность связывающих молекул, таких как клетки, экспрессирующие связывающие молекулы, например TCR или CAR, вызывать иммунный ответ, может быть определена путем измерения высвобождения цитокинов. В некоторых вариантах осуществления в ответ на совместное культивирование или экспонирование с клетками, экспрессирующими связывающие молекулы, например, TCR или CAR, различные цитокины высвобождаются, когда клетки стимулируются соответствующей клеткой-мишенью, которая, как известно, экспрессирует HPV 16, такой как HPV 16 Е6 или HPV 16 Е7. Неограничивающие примеры таких цитокинов включают IFN-γ, TNF-α и GM-CSF. Иллюстративные клетки, которые, как известно, экспрессируют HPV 16, включают, но не ограничиваются ими, клетки CaSki (ATCC No. CRL-1550, которые содержат примерно 600 копий интегрированного HPV16) или другие опухолевые клетки, экспрессирующие соответствующую молекулу MHC и соответствующий пептидный эпитоп, например эпитоп E6 или E7 HPV 16, такой как любой из представленных в SEQ ID NO: 232 -239.
[0458] В некоторых вариантах осуществления может быть определена активация CTL. Существует множество методов для анализа активности CTL. В некоторых вариантах осуществления активность CTL можно оценить, анализируя культуру на наличие CTL, которые лизируют радиоактивно меченные клетки-мишени, такие как мишени, стимулированные специфическим пептидом. Эти методы включают мечение клеток-мишеней радионуклидами, такими как Na2, 51CrO4 или 3H-тимидином, и измерение высвобождения или удержания радионуклидов в клетках-мишенях в качестве показателя клеточной смерти. В некоторых вариантах осуществления известно, что CTL высвобождают различные цитокины, когда они стимулируются соответствующей клеткой-мишенью, такой как опухолевая клетка, экспрессирующая соответствующую молекулу МНС и соответствующий пептидный эпитоп, и присутствие таких эпитоп-специфичных CTL может определяется путем измерения высвобождения цитокинов. Неограничивающие примеры таких цитокинов включают IFN-γ, TNF-α и GM-CSF. Анализы на эти цитокины хорошо известны в данной области, и их выбор оставлен на усмотрение специалиста. Методология измерения как клеточной смрти клеток-мишеней, так и высвобождения цитокинов в качестве меры реактивности CTL приведена в Coligan, J.E. et al. (Current Protocols in Immunology, 1999, John Wiley & Sons, Inc., Нью-Йорк).
[0459] В некоторых вариантах осуществления продуцирование цитокинов может быть измерено как показатель иммунного ответа. В некоторых случаях такие измеренные цитокины могут включать, без ограничения, интерлейкин-2 (IL-2), интерферон-гамма (IFNγ), интерлейкин-4 (IL-4), TNF-альфа, интерлейкин-6 (IL-6), интерлейкин-10 (IL-10), интерлейкин-12 (IL-12) или TGF-бета. Анализы для измерения цитокинов хорошо известны в данной области и включают, без ограничения, ИФА, внутриклеточное окрашивание цитокинов, тест-систему для определния цитокинов (cytometric bead array), ОТ-ПЦР, ELISPOT, проточную цитометрию и биологические анализы, в которых клетки, чувствительные к соответствующему цитокину, тестируют на респонсивность (например, пролиферацию) в присутствии тестируемого образца.
[0460] В некоторых вариантах осуществления клетки, котоорые подвергаются воздействию связывающих молекул, например клетки, содержащие связывающие молекулы, такие как TCR или CAR, оценивают на иммунологическое считывание, такое как Т-клеточный анализ. В некоторых вариантах осуществления клетки, содержащие связывающую молекулу, могут активировать ответ CD8+ T-клеток. В одном варианте осуществления ответы CD8+ Т-клеток можно оценивать путем мониторинга реактивности CTL с использованием анализов, которые включают, но не ограничиваются ими, лизис клеток-мишеней через высвобождение 51Cr или детектирование высвобождения интерферона-гамма, например, с помощью твердофазного иммуноферментного анализа (ИФА), внутриклеточного окрашивания цитокинов или ELISPOT. В некоторых вариантах осуществления связывающие молекулы, например клетки, содержащие связывающие молекулы, такие как TCR или CAR, могут активировать ответ CD4+ T-клеток. В некоторых аспектах ответы CD4+ T-клеток можно оценивать с помощью анализов, которые измеряют пролиферацию, таких как включение [3H] -тимидина в клеточную ДНК и/или продуцирование цитокинов, таких как ИФА, внутриклеточное окрашивание цитокинов или ELISPOT. В некоторых случаях цитокин может включать, например, интерлейкин-2 (IL-2), интерферон-гамма (IFN-гамма), интерлейкин-4 (IL-4), TNF-альфа, интерлейкин-6 (IL-6), интерлейкин-10 (IL-10), интерлейкин-12 (IL-12) или TGF бета. В некоторых вариантах осуществления распознавание или связывание пептидного эпитопа, такого как эпитоп МНС класса II, с помощью связывающей молекулы может вызывать или активировать ответ CD4+ T-клеток и/или ответ CD8+ T-клеток.
[0461] В некоторых вариантах осуществления специфичность и/или функция связывания (например, способность вызывать иммунный ответ на HPV 16) связывающей молекулы, например, TCR или ее антигенсвязывающего фрагмента, является, по меньшей мере, частично CD8-независимой. В некоторых случаях распознавание TCR пептида в контексте молекулы МНС и последующая активация Т-клеток облегчаются в присутствии корецептора CD8. Например, взаимодействие с корецептором CD8 может облегчать низкоаффинные или умеренноаффинные взаимодействиям TCR и/или активацию T-клеток (см., например, Kerry et al. J. Immunology (2003) 171 (9): 4493-4503 и Robbins et al. J. Immunology (2008) 180 (9): 6116-6131). Среди представленных связывающих молекул есть молекулы, например, TCR, которые демонстрируют CD8-независимое связывание с пептидным эпитопом E6 или E7 HPV. В некоторых вариантах осуществления такие связывающие молекулы, например, TCR могут иметь более высокую функциональную авидность или аффинность, чем TCR или их антигенсвязывающие фрагменты, которые требуют наличия коэкспрессии CD8. В некоторых аспектах представленные CD8-независимые связывающие молекулы, такие как TCR, могут быть экспрессированы или сконструированы в клетках, например, Т-клетках, которые не экспрессируют CD8, как например, могут быть экспрессированы или сконструированы в клетках CD4+. В некоторых вариантах осуществления среди представленных сконструированных клеток, не экспрессирующих CD8, например, CD4+ клетки представляют собой клетки, экспрессирующие рекомбинантную связывающую молекулу, например, TCR или антигенсвязывающий фрагмент, которые демонстрируют, по меньшей мере, или, по меньшей мере, примерно 20%, 30%, 40%, 50%, 60%, 70%, 80%, 90% или более специфичности связывания, аффинности и/или авидности к пептиду в контексте молекулы МНС в виде той же самой связывающей молекулы (например, TCR или ее антигенсвязывающего фрагмента), которая экспрессируется на CD8+ Т-клетке.
II. Нуклеиновые кислоты, векторы и способы экспрессии
[0462] Также представлены нуклеиновые кислоты, кодирующие любую из представленных связывающих молекул, например TCR или их антигенсвязывающие фрагменты, или антитела или их антигенсвязывающие фрагменты, или CAR, содержащие такие антитела, такие как те, которые описаны в настоящем документе. Нуклеиновые кислоты могут включать те, которые включают природные и/или не встречающиеся в природе нуклеотиды и основания, например, такие, которые содержат модификации остова. Термины «молекула нуклеиновой кислоты», «нуклеиновая кислота» и «полинуклеотид» могут использоваться взаимозаменяемо и относятся к полимеру нуклеотидов. Такие полимеры нуклеотидов могут содержать природные и/или неприродные нуклеотиды и включают, но не ограничиваются ими, ДНК, РНК и ПНК. «Последовательность нуклеиновой кислоты» относится к линейной последовательности нуклеотидов, которые включают молекулу нуклеиновой кислоты или полинуклеотид.
[0463] В некоторых вариантах осуществления связывающая молекула, например TCR или его антигенсвязывающая часть могут представлять собой природный белок, полученный рекомбинантным путем, или его мутированную форму, в которой одно или более свойств, таких как характеристика связывания, были изменены. В некоторых аспектах нуклеиновая кислота является синтетической. В некоторых случаях нуклеиновая кислота представляет собой или содержит кДНК. В некоторых аспектах молекула нуклеиновой кислоты может быть модифицирована для использования в конструкциях, описанных в настоящем документе, как например, для оптимизации кодонов. В некоторых случаях последовательности могут быть сконструированы так, чтобы содержать концевые последовательности сайтов рестрикции для целей клонирования в векторы.
[0464] В некоторых вариантах осуществления молекула нуклеиновой кислоты, кодирующая связывающую молекулу, например TCR, может быть получена из различных источников, как например с помощью полимеразной цепной реакции амплификации (ПЦР) кодирующих нуклеиновых кислот внутри данной клетки или клеток или выделенных из них. В некоторых вариантах осуществления TCR получают из биологического источника, такого как клетки, такие как T-клетки (например, цитотоксические T-клетки), гибридомы T-клеток или другие общедоступные источники. В некоторых вариантах осуществления TCR может быть получен от одного из различных видов животных, таких как человек, мышь, крыса или другое млекопитающее, как например, обычно от человека. В некоторых вариантах осуществления Т-клетки могут быть получены из выделенных клеток in vivo, таких как клетки нормальных (или здоровых) индивидуумов или больных пациентов, включая Т-клетки, присутствующие в мононуклеарных клетках периферической крови (РВМС) или инфильтрирующие опухоль лимфоциты (TIL). В некоторых вариантах осуществления Т-клетки могут представлять собой культивируемые гибридомы Т-клеток или клон. Например, в некоторых вариантах осуществления для создания вектора, кодирующего TCR, α- и β-цепи можно амплифицировать с помощью ПЦР из общей кДНК, выделенной из клона Т-клеток, экспрессирующих интересующий TCR, и клонировать в экспрессирующий вектор. В некоторых вариантах осуществления α- и β-цепи могут быть получены синтетически. В некоторых вариантах осуществления α- и β-цепи клонируются в один и тот же вектор.
[0465] В некоторых вариантах осуществления TCR или его антигенсвязывающая часть могут быть получены синтетически на основе знания последовательности TCR.
[0466] В некоторых вариантах осуществления молекула нуклеиновой кислоты содержит последовательность нуклеиновой кислоты, кодирующую альфа-цепь, и/или нуклеотидную последовательность, кодирующую бета-цепь.
[0467] В некоторых вариантах осуществления последовательность нуклеиновой кислоты, кодирующая альфа-цепь, содержит одно из следующего: остатки 61-816 SEQ ID NO: 20, остатки 58-804 SEQ ID NO: 30, остатки 61-825 SEQ ID NO: 40, остатки 64-813 SEQ ID NO: 50, остатки 64-816 SEQ ID NO: 60, остатки 58-807 SEQ ID NO: 70, остатки 61-825 SEQ ID NO: 80, остатки 67-831 SEQ ID NO: 90, остатки 58-801 SEQ ID NO: 100, остатки 64-810 SEQ ID NO: 183, остатки 58-801 SEQ ID NO: 202, остатки 67-813 SEQ ID NO: 219, их вырожденную последовательность или последовательность, имеющую с ними по меньшей мере 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичности последовательности. В некоторых аспектах нуклеотидная последовательность, кодирующая бета-цепь, содержит одно из следующего: остатки 58-936 SEQ ID NO: 17, остатки 58-930 SEQ ID NO: 16, остатки 58-939 SEQ ID NO: 24, остатки 64-930 SEQ ID NO: 34 или 44, остатки 58-933 SEQ ID NO: 55, остатки 58-927 SEQ ID NO: 64, остатки 64-936 SEQ ID NO: 74, остатки 58-933 SEQ ID NO: 84, остатки 63-930 из SEQ ID NO: 94, остатки 46-936 SEQ ID NO: 104, остатки 58-933 SEQ ID NO: 108, их вырожденную последовательность или последовательность, имеющую с ними по меньшей мере 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичности последовательности.
[0468] В некоторых вариантах осуществления нуклеотидная последовательность, кодирующая альфа-цепь, и/или нуклеотидная последовательность, кодирующая бета-цепь, оптимизирована по кодонам. Как правило, оптимизация кодонов включает баланс процентного соотношения кодонов, отобранных с использованием известной представленности транспортной РНК человека, так что ни одна из них не перегружена или не ограничена. Это может быть необходимо в некоторых случаях, потому что большинство аминокислот кодируются более чем одним кодоном, и использование кодонов варьируется от организма к организму. Различия в использовании кодонов между трансфецированными генами и клетками-хозяевами могут влиять на экспрессию белка и иммуногенность конструкции нуклеиновой кислоты. В общем, для оптимизации кодонов выбираются кодоны для отбора таких кодонов, которые находятся в балансе с частотой их использования у человека. Как правило, избыточность кодонов для аминокислот такова, что разные кодоны кодируют одну аминокислоту. В некоторых вариантах осуществления при выборе кодона для замены может быть желательно, чтобы получающаяся мутация представляла собой молчащую мутацию, так чтобы замена кодона не влияла на аминокислотную последовательность. Как правило, последний нуклеотид кодона может оставаться неизменным, не влияя на аминокислотную последовательность.
[0469] В некоторых случаях последовательность нуклеиновой кислоты, кодирующая альфа-цепь, содержит одно из следующего: остатки 67-825 SEQ ID NO: 10, остатки 58-813 SEQ ID NO: 11, остатки 64-822 SEQ ID NO: 12 остатки 61-825 SEQ ID NO: 21, остатки 58-813 SEQ ID NO: 31, остатки 61-834 SEQ ID NO: 41, остатки 63-822 SEQ ID NO: 51, остатки 64-825 SEQ ID NO: 61, остатки 58-816 SEQ ID NO: 71, остатки 61-834 SEQ ID NO: 81, остатки 67-840 SEQ ID NO: 91, остатки 58-810 SEQ ID NO: 101, их вырожденную последовательность или последовательность, имеющую с ними по меньшей мере 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичности последовательности. В некоторых примерах нуклеотидная последовательность, кодирующая бета-цепь, содержит одно из следующего: остатки 58-930 SEQ ID NO: 7, остатки 58-936 SEQ ID NO: 8, остатки 58-933 SEQ ID NO: 9 остатки 58 -939 SEQ ID NO: 25, остатки 64-930 SEQ ID NO: 35, 45 или 95, остатки 58-933 SEQ ID NO: 54 или 85, остатки 58-927 SEQ ID NO: 65, остатки 64-936 SEQ ID NO: 75, остатки 46-936 SEQ ID NO: 105, их вырожденную последовательность или последовательность, имеющая с ними по меньшей мере 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичности последовательности.
[0470] В некоторых вариантах осуществления молекула нуклеиновой кислоты, кодирующая альфа-цепь и/или бета-цепь TCR, содержит последовательность нуклеиновой кислоты, соответствующую SEQ ID NO, представленным в Таблице 11. Также среди представленных молекул нуклеиновой кислоты, кодирующих TCR, имеются такие, которые содержат последовательности, по меньшей мере или примерно на 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99%, идентичные таким последовательностям. Иллюстративные TCR, кодированные такими последовательностями, или их модифицированные версии, также представлены в Таблице 11.
[0471] Также представлены векторы или конструкции, содержащие такие молекулы нуклеиновой кислоты. В некоторых вариантах осуществления векторы или конструкции содержат один или более промоторов, функционально связанных с нуклеотидом, кодирующим альфа-цепь и/или бета-цепь. В некоторых вариантах осуществления промотор функционально связан с одной или болееи молекулами нуклеиновой кислоты.
[0472] В некоторых вариантах осуществления вектор или конструкция могут содержать один промотор, который управляет экспрессией одной или более молекул нуклеиновой кислоты. В некоторых вариантах осуществления такие промоторы могут быть полицистронными (бицистронными или трицистронными, см., например, Патент США № 6060273). Например, в некоторых вариантах осуществления транскрипционные единицы могут быть сконструированы как бицистронные единицы, содержащие IRES (внутренний участок посадки рибосомы), который позволяет коэкспрессировать генные продукты (например, кодировать альфа-цепь и/или бета-цепь TCR) посредством сообщения от одного промотора. Альтернативно, в некоторых случаях один промотор может направлять экспрессию РНК, которая содержит в одной открытой рамке считывания (ORF) два или три гена (например, кодирующих альфа-цепь и/или бета-цепь TCR), отделенных друг от друга последовательностями, кодирующими саморасщепляемый пептид (например, T2A) или сайт узнавания протеазы (например, фурина). Таким образом, ORF кодирует один полипротеин, который либо во время трансляции (в случае 2A, например, T2A), либо после трансляции расщепляется на отдельные белки. В некоторых случаях пептид, такой как T2A, может заставить рибосому пропускать (пропуск рибосомы) синтез пептидной связи на C-конце элемента 2A, приводя к разделению между концом последовательности 2A и следующим пептидом, расположенным в 3'-области (см., например, de Felipe. Genetic Vaccines and Ther. с2:13 (2004) и deFelipe et al. Traffic 5: 616-626 (2004). Примерами пептидов расщепления 2А, включая те, которые могут индуцировать пропуск рибосомы, являются вирус Thosea asigna (T2A, например, SEQ ID NO: 211). или 274), свиной тешовирус-1 (P2A, например, SEQ ID NO: 204 или 345), вирус конского ринита A (E2A, например, SEQ ID NO: 346) и последовательности 2A из вируса ящура (F2A, например, SEQ ID NO: 344), как описано в Патентной публикации США № 2007/0116690.
[0473] В некоторых случаях нуклеотидная последовательность, кодирующая альфа-цепь, и нуклеотидная последовательность, кодирующая бета-цепь, разделены нуклеотидной последовательностью, кодирующей внутренний участок посадки рибосомы (IRES), или пептидной последовательностью, которая вызывает пропуск рибосомы. В некоторых случаях нуклеотидная последовательность, кодирующая альфа-цепь, и нуклеотидная последовательность, кодирующая бета-цепь, разделены пептидной последовательностью, которая вызывает пропуск рибосомы. В некоторых таких случаях пептид, который вызывает пропуск рибосомы, представляет собой пептид P2A или T2A и/или содержит последовательность аминокислот, представленную в SEQ ID NO: 204, 211, 274 или 345. В некоторых аспектах нуклеотидная последовательность, кодирующая пептид, который вызывает пропуск рибосомы, содержит последовательность, представленную в SEQ ID NO: 4, 5, 6, 207, 208, 209 или 210, 347, 1096, 1179, 1180 или 1181.
[0474] В некоторых вариантах осуществления последовательность нуклеиновой кислоты, кодирующая альфа-цепь, и нуклеотидная последовательность, кодирующая бета-цепь, присутствуют в любом порядке, разделенные нуклеотидной последовательностью, кодирующей внутренний участок посадки рибосомы (IRES), или пептидной последовательностью, которая вызывает пропуск рибосомы. Например, в некоторых вариантах осуществления молекула нуклеиновой кислоты содержит последовательность нуклеиновой кислоты, кодирующую бета-цепь, последовательность нуклеиновой кислоты, кодирующую последовательность IRES или пептид, который вызывает пропуск рибосомы, например последовательность P2A или T2A, как описано в настоящем документе, и нуклеиновую последовательность нуклеиновой кислоты, которая кодирует альфа-цепь, в таком порядке. В других вариантах осуществления молекула нуклеиновой кислоты содержит последовательность нуклеиновой кислоты, которая кодирует альфа-цепь, последовательность нуклеиновой кислоты, которая кодирует IRES, или пептидную последовательность, которая вызывает пропуск рибосомы, и последовательность нуклеиновой кислоты, которая кодирует бета-цепь, в таком порядке.
[0475] Таким образом, в некоторых аспектах молекула нуклеиновой кислоты кодирует полипептид, содержащий бета-цепь, IRES или пептид, который вызывает пропуск рибосомы, и альфа-цепь, в таком порядке. В других аспектах молекула нуклеиновой кислоты кодирует полипептид, содержащий альфа-цепь, IRES или пептид, который вызывает пропуск рибосомы, и бета-цепь, в таком порядке.
[0476] В некоторых вариантах осуществления молекула нуклеиновой кислоты кодирует полипептид, содержащий аминокислотную последовательность, представленную в Таблице 12, или последовательность, имеющую с ними по меньшей мере, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% идентичности последовательности. В некоторых вариантах осуществления молекула нуклеиновой кислоты кодирует полипептид, представленный в любой из SEQ ID NO: 1, 2, 3, 27, 37, 47, 57, 67, 77, 87, 97, 107, 223, 224, 225, 226, 227, 228, 229, 230,231, 340-342, 350-388, 391-429 или 1383-1384, или последовательности, имеющей с ними по меньшей мере 90%, 91%, 92%, 93%, 94%, 95% 96%, 97%, 98% или 99% идентичности последовательности. В некоторых вариантах осуществления молекула нуклеиновой кислоты содержит последовательность нуклеиновой кислоты, представленную в любой из SEQ ID NO: 13, 14, 15, 26, 36, 46, 56, 66, 76, 86, 96, 106, 432-472, или 1382, или последовательность, имеющую с ними по меньшей мере 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% идентичности последовательности.
[0477] Также представлены полипептиды, содержащие последовательность, кодируемую любой из представленных нуклеиновых кислот. В некоторых аспектах полипептид содержит аминокислотную последовательность, соответствующую SEQ ID NO, представленным в Таблице 12, или последовательность, имеющую с ними по меньшей мере 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% идентичности последовательности. В некоторых вариантах осуществления полипептид содержит последовательность, представленную в любой из SEQ ID NO: 1, 2, 3, 27, 37, 47, 57, 67, 77, 87, 97, 107, 223, 224, 225, 226, 227., 228, 229, 230, 231, 340-342, 350-388, или 391-429, или 1383-1384, или последовательность, имеющую с ними, по меньшей мере, 90%, 91%, 92%, 93%, 94%, 95% 96%, 97%, 98% или 99% идентичности последовательности. Примеры таких TCR или их модифицированных версий также представлены в Таблице 12.
[0478] В некоторых вариантах осуществления молекула нуклеиновой кислоты может дополнительно кодировать маркер (например, EGFRt или другой маркер, как описано), который отделен от CAR или отделен от цепей TCR с помощью линкера, такого как расщепляемая линкерная последовательность или пептидная последовательность, которая вызывает пропуск рибосомы, например, T2A или P2A.
[0479] В некоторых вариантах осуществления конструкция может быть расположена в любом порядке, так что последовательность, кодирующая маркер, находится в 3'-области относительно последовательности альфа и/или бета, в 5'-области относительно последовательности альфа и/или бета и/или между последовательностью альфа и бета, где в некоторых случаях каждый отдельный компонент разделен расщепляемой линкерной последовательностью или пептидом, который вызывает пропуск рибосомы (например, T2A или P2A) или IRES. В некоторых вариантах осуществления молекула нуклеиновой кислоты содержит в указанном порядке последовательность нуклеиновой кислоты, которая кодирует маркер (например, EGFRt), расщепляемый линкер или последовательность пропуска рибосомы (например, T2A или P2A), бета-цепь, расщепляемый линкер или последовательность пропуска рибосомы (например, T2A или P2A) и альфа-цепь. В некоторых вариантах осуществления молекула нуклеиновой кислоты содержит в указанном порядке последовательность нуклеиновой кислоты, которая кодирует маркер (например, EGFRt), расщепляемый линкер или последовательность пропуска рибосомы (например, T2A или P2A), альфа-цепь, расщепляемый линкер или последовательность пропуска рибосомы (например, T2A или P2A) и бета-цепь. В некоторых вариантах осуществления молекула нуклеиновой кислоты содержит в указанном порядке последовательность нуклеиновой кислоты, которая кодирует бета-цепь, расщепляемый линкер или последовательность пропуска рибосом (например, T2A или P2A), альфа-цепь, расщепляемый линкер или последовательность пропуска рибосом (например, T2A или P2A) и маркер (например, EGFRt). В некоторых вариантах осуществления молекула нуклеиновой кислоты содержит в указанном порядке последовательность нуклеиновой кислоты, которая кодирует альфа-цепь, расщепляемый линкер или последовательность пропуска рибосом (например, T2A или P2A), бета-цепь, расщепляемый линкер или последовательность пропуска рибосом (например, T2A или P2A) и маркер (например, EGFRt). В некоторых вариантах осуществления молекула нуклеиновой кислоты содержит в указанном порядке последовательность нуклеиновой кислоты, которая кодирует альфа-цепь, расщепляемый линкер или последовательность пропуска рибосомы (например, T2A или P2A), маркер (например, EGFRt), расщепляемый линкер или последовательность пропуска рибосомы (например, T2A или P2A) и бета-цепь. В некоторых вариантах осуществления молекула нуклеиновой кислоты содержит в указанном порядке последовательность нуклеиновой кислоты, которая кодирует бета-цепь, расщепляемый линкер или последовательность пропуска рибосомы (например, T2A или P2A), маркер (например, EGFRt), расщепляемый линкер или последовательность пропуска рибосомы (например, T2A или P2A) и альфа-цепь.
[0480] В некоторых вариантах осуществления введение конструкции, кодирующей CAR и EGFRt, разделенных переключателем рибосомы T2A, может экспрессировать два белка из одной и той же конструкции, так что EGFRt можно использовать в качестве маркера для детектирования клеток, экспрессирующих такую конструкцию.
[0481] Нуклеиновая кислота может кодировать аминокислотную последовательность, содержащую вариабельную область альфа (Vα) или вариабельную область легкой цепи (VL) TCR или антитела, соответственно. В некоторых случаях нуклеиновая кислота кодирует аминокислотную последовательность, содержащую вариабельную область бета (Vβ) или вариабельную область тяжелой цепи (VH) TCR или антитела, соответственно. В дополнительном варианте осуществления представлены один или более векторов (например, экспрессирующих векторов), содержащих такую нуклеиновую кислоту.
[0482] Также представлены векторы, такие как те, которые содержат любую из нуклеиновых кислот, описанных в настоящем документе. В некоторых вариантах осуществления нуклеиновую кислоту или нуклеиновые кислоты, кодирующие одну или обе цепи связывающей молекулы, например TCR, клонируют в подходящий вектор или экспрессирующие векторы. Экспрессирующий вектор может быть любым подходящим рекомбинантным экспрессирующим вектором и может использоваться для трансформации или трансфекции любого подходящего хозяина. Подходящие векторы включают векторы, предназначенные для размножения и наращивания или для экспрессии, или того и другого, такие как плазмиды и вирусы. В некоторых вариантах осуществления вектор представляет собой экспрессирующий вектор.
[0483] В некоторых вариантах осуществления вектор может представлять собой вектор серии pUC (Fermentas Life Sciences), серии pBluescript (Stratagene, Ла Холья, Калифорния), серии pET (Novagen, Мэдисон, Висконсин), Серии pGEX (Pharmacia Biotech, Упсала, Швеция) или серия pEX (Clontech, Пало-Альто, Калифорния). В некоторых случаях также могут использоваться векторы бактериофагов, такие как λG10, λGT11, λZapII (Stratagene), λEMBL4 и λNM1149. В некоторых вариантах осуществления могут использоваться экспрессирующие векторы растений, и они включают pBI01, pBI101.2, pBI101.3, pBI121 и pBIN19 (Clontech). В некоторых вариантах осуществления экспрессирующие векторы животных включают pEUK-Cl, pMAM и pMAMneo (Clontech). В некоторых случаях вектор является вирусным вектором. В некоторых таких аспектах вирусный вектор представляет собой ретровирусный вектор, такой как лентивирусный вектор. векторы экспрессииВ некоторых случаях лентивирусный вектор происходит из HIV-1.
[0484] В некоторых вариантах осуществления рекомбинантные экспрессирующие векторы могут быть получены с использованием стандартных методов рекомбинантной ДНК. В некоторых вариантах осуществления векторы могут содержать регуляторные последовательности, такие как кодоны инициации и терминации транскрипции и трансляции, которые специфичны для типа хозяина (например, бактерии, гриба, растения или животного), в который должен быть введен вектор, если это целесообразно, и принимая во внимание, является ли вектор на основе ДНК или РНК. В некоторых вариантах осуществления вектор может содержать ненативный промотор, функционально связанный с нуклеотидной последовательностью, кодирующей связывающую молекулу, такую как TCR, антитело или его антигенсвязывающий фрагмент. В некоторых вариантах осуществления промотор может представлять собой невирусный промотор или вирусный промотор, такой как промотор цитомегаловируса (CMV), промотор SV40, промотор RSV и промотор, присутствующий в длинном концевом повторе вируса мышиных стволовых клеток. Другие промоторы, известные специалисту в данной области, также рассматриваются.
[0485] Также представлены способы получения связывающих молекул (включая антигенсвязывающие фрагменты). В некоторых вариантах осуществления представлена клетка-хозяин, содержащая такую нуклеиновую кислоту. Для рекомбинантной продукции связывающих молекул нуклеиновая кислота, кодирующая связывающую молекулу, например, как описано выше, может быть выделена и вставлена в один или более векторов для дальнейшего клонирования и/или экспрессии в клетке-хозяине. Такая нуклеиновая кислота может быть легко выделена и секвенирована с использованием традиционных процедур (например, с использованием олигонуклеотидных зондов, которые способны специфически связываться с генами, кодирующими альфа- и бета-цепи TCR или тяжелые и легкие цепи антитела). В некоторых вариантах осуществления предложен способ получения связывающей молекулы, где способ включает культивирование клетки-хозяина, содержащей нуклеиновую кислоту, кодирующую связывающую молекулу, как указано выше, в условиях, подходящих для экспрессии связывающей молекулы, и, возможно, восстановление связывающей молекулы из клетки-хозяина (или культуральной среды клетки-хозяина).
[0486] В одном таком варианте осуществления клетка-хозяин содержит (например, была трансформирована) вектор, содержащий нуклеиновую кислоту, которая кодирует аминокислотную последовательность, содержащую область Vβ TCR или его антигенсвязывающий фрагмент, и нуклеиновую кислоту, которая кодирует аминокислотную последовательность, содержащую область Vα TCR или его антигенсвязывающий фрагмент. В другом таком варианте осуществления клетка-хозяин содержит (например, была трансформирована) вектор, содержащий нуклеиновую кислоту, которая кодирует аминокислотную последовательность, содержащую VH антитела или его антигенсвязывающего фрагмента и VL антитела или антигенсвязывающего его фрагмента. В некоторых аспектах клетка-хозяин содержит (например, была трансформирована): первый вектор, содержащий нуклеиновую кислоту, которая кодирует аминокислотную последовательность, содержащую область Vα TCR или его антигенсвязывающего фрагмента, и второй вектор, содержащий нуклеиновую кислоту, кодирующая аминокислотную последовательность, содержащую область Vβ TCR или его антигенсвязывающего фрагмента. В других аспектах клетка-хозяин содержит (например, была трансформирована): первый вектор, содержащий нуклеиновую кислоту, которая кодирует аминокислотную последовательность, содержащую VL антитела или его антигенсвязывающий фрагмент, и второй вектор, содержащий нуклеиновую кислоту, которая кодирует аминокислотную последовательность, содержащую VH антитела или его антигенсвязывающий фрагмент.
[0487] В дополнение к прокариотам эукариотические микробы, такие как нитчатые грибы или дрожжи, являются подходящими хозяевами для клонирования или экспрессии для векторов, кодирующих связывающие молекулы, включая грибы и штаммы дрожжей, пути гликозилирования которых были модифицированы, чтобы имитировать или приблизить их к клеткам человека. Смотри Gerngross, Nat. Biotech. 22: 1409-1414 (2004) и Li et al., Nat. Biotech. 24: 210-215 (2006).
[0488] Иллюстративные эукариотические клетки, которые можно использовать для экспрессии полипептидов, включают, но не ограничиваются ими, клетки COS, включая клетки COS 7; клетки 293, в том числе клетки 293-6E; клетки CHO, включая CHO-S, DG44. Клетки Lec13 CHO и клетки FUT8 СНО; Клетки PER.C6®; и клетки NSO. В некоторых вариантах осуществления конкретная эукариотическая клетка-хозяин выбирается на основании ее способности осуществлять желаемые посттрансляционные модификации в связывающей молекуле. Например, в некоторых вариантах осуществления клетки СНО продуцируют полипептиды, которые имеют более высокий уровень сиалилирования, чем тот же полипептид, продуцируемый в клетках 293. В некоторых вариантах осуществления связывающая молекула продуцируется в бесклеточной системе. Иллюстративные бесклеточные системы описаны, например, в Sitaraman et al., Methods Mol. Biol. 498: 229-44 (2009); Spirin, Trends Biotechnol. 22: 538-45 (2004); Endo et al., Biotechnol. Adv. 21: 695-713 (2003).
III. Методы идентификации и генерации Т-клеточных рецепторов
[0489] В некоторых вариантах осуществления предложены способы идентификации и генерирования Т-клеточных рецепторов, направленных на антиген-мишень. В некоторых аспектах способы включают воздействие на биологические образцы, содержащие Т-клетки, такие как первичные Т-клетки, включая образцы, полученные от нормальных доноров или пациентов, имеющих заболевание или состояние, представляющее интерес, множественных раундов воздействия антигена и оценки. В некоторых аспектах раунды включают использование искусственных или сконструированных антигенпрезентирующих клеток, таких как аутологичные дендритные клетки или другие APC, стимулированные желаемым пептидным антигеном, для стимулирования презентирования на MHC, таком как MHC класса I или II. В некоторых аспектах проводят множественные раунды воздействия антигена, а в некоторых аспектах Т-клетки сортируют после одного или более раундов, например, на основании способности связываться с желаемым антигеном (таким как тетрамеры пептид-МНС). В некоторых аспектах сортировка осуществляется проточной цитометрией. В некоторых аспектах клетки из клеток, которые, как считается, связываются с желаемым антигеном (положительная фракция), и клетки, которые, как считается, не связываются с антигеном, оценивают, например, методами секвенирования отдельных клеток. В некоторых аспектах метод определяет последовательности и идентифицирует на уровне одной клетки TCR, присутствующие в каждом образце. В некоторых аспектах способы могут количественно определять количество копий данной пары TCR, присутствующих в образце, и, как таковые, могут оценивать количество данного TCR в данном образце и/или его обогащение по сравнению с другим образцом, например обогащение или избыток в положительной (антигенсвязывающей) фракции, например, в течение одного или более раундов, например, по сравнению с отрицательной фракцией. В некоторых аспектах такие анализы проводят для генерирования антигенспецифических Т-клеточных рецепторов (TCR), которые специфически связываются с пептидными антигенами вируса папилломы человека 16 или 18, такими как пептиды, полученные из E6 или E7, такие как E6 (29-38) или E7 (11-19) пептид, например, презентированный на молекулах MHC-I и выживший и/или обогащенный в течение некоторого времени после множества раундов стимуляции антигеном. В некоторых аспектах создаются клональные Т-клеточные линии, и последовательности отдельных спаренных альфа- и бета-цепей TCR и их содержание в различных популяциях определяют на основе одной клетки с использованием высокопроизводительного секвенирования спаренного TCR.
[0490] В некоторых аспектах пептид-стимулированные HLA: A02: 01APC были получены с использованием пептида E6 HPV 16 (29-38) (TIHDIILECV; SEQ ID NO: 233) или пептида E7 (11-19) (YMLDLQPET; SEQ ID NO: 236). Аутологичные CD8+ Т-клетки от нормальных человеческих доноров инкубируют в течение нескольких циклов с пептид-стимулированными клетками, и отбор проводят на основе связывания аутологичными тетрамерами МНС с пептидной нагрузкой.
[0491] В некоторых аспектах клетки подвергали множественным, таким как в общей сложности двум, трем или более, рацндам стимуляции в присутствии пептид-стимулированных клеток (например, с определенной концентрацией пептида 1000 нг/мл, поддерживаемой в течение трех раундов). После одного или более из таких раундов, как, например, после первого и/или после второго и третьего раундов стимуляции, клетки сортировали с помощью проточной цитометрии на популяции, положительные и отрицательные, соответственно, для связывания с тетрамерами пептид-МНС, содержащими соответствующий тетрамер. Клетки тетрамер-положительных и отрицательных популяций, после каждого или одного или более из одного или более, как например после второго и третьего, раундов в некоторых аспектах, подвергают секвенированию TCR из одной клетки, чтобы оценить наличие и частоту встречаемости отдельных TCR в разных популяциях, а также устойчивость клонов TCR в течение множества раундов стимуляции антигеном.
[0492] В некоторых аспектах клеточные популяции из положительных и отрицательных фракций (то есть отсортированных методом проточной цитометрии на основе положительного и отрицательного окрашивания соответственно для связывания с соответствующим антигеном, таким как пептид-MHC, таким как тетрамеры с нагрузкой, например, как определено проточной цитометрией), после одного или более раундов, подвергаются высокопроизводительному секвенированию одной клетки для пар альфа- и бета-цепей TCR. Секвенирование TCR с высокой призводительностью в одной клетке в некоторых аспектах выполняется, как в целом описано в опубликованных патентных заявках PCT, публикациях под номерами WO2012/048340, WO2012/048341 и WO2016/044227. Таким образом, в методах секвенирования в некоторых аспектах используются капли, содержащие одну клетку, и образцы и молекулярные штрих-коды, чтобы идентифицировать отдельные пары последовательностей альфа- и бета-цепей TCR на уровне одной клетки для каждой из большого числа (например, миллионов) отдельных клеток, присутствующих в одной исходной композиции, и оценить численность каждой пары TCR в различных оцененных популяциях. Способность идентифицировать и количественно определять пары TCR на уровне одной клетки в некоторых вариантах осуществления позволяет оценивать частоту каждой из различных пар TCR в каждой из отдельных положительных и отрицательных фракций, а также оценивать обогащение и стойкость TCR в течение множества раундов антигенной стимуляции.
[0493] В некоторых аспектах способы генерируют, идентифицируют, выделяют и/или отбирают пары TCR, которые обогащены антигенсвязывающим, например, пептид-связывающими, фракциями, после по меньшей мере одного, а в некоторых аспектах - после множества раундов стимуляции. В некоторых аспектах TCR присутствуют и/или присутствуют в желаемом количестве и/или предпочтительно обогащены после раундов 1, 2 и/или 3, а в некоторых аспектах, по меньшей мере, после множества раундов воздействия антигена. В некоторых аспектах TCR обогащаются в популяции с течением времени после нескольких циклов воздействия антигена. Также предложены TCR, полученные или идентифицированные с использованием таких способов, такие как TCR, обладающие такими свойствами, как способность выживать и/или распространяться в течение нескольких раундов воздействия антигена, например, в анализе пептид-стимулированных АРС.
IV. Сконструированные клетки
[0494] Также предложены клетки, такие как клетки, которые были сконструированы так, чтобы содержать связывающую молекулу, описанную в настоящем документе. Также предложены популяции таких клеток, композиции, содержащие такие клетки и/или обогащенные такими клетками, как например, в которых клетки, экспрессирующие связывающую молекулу, составляют, по меньшей мере, 15%, 20%, 25%, 30%, 35%, 40%., 50%, 60%, 70%, 80%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более процентов от общего числа клеток в композиции или клетках определенного типа, таких как Т-клетки или CD8+ или CD4+ клетки. В некоторых вариантах осуществления клетки являются первичными Т-клетками. Среди композиций представлены фармацевтические композиции и составы для введения, например, для адоптивной клеточной терапии. Также предложены терапевтические способы введения клеток и композиций индивидуумам, например пациентам.
[0495] Таким образом, также предложены генетически сконструированные клетки, экспрессирующие связывающие молекулы. Клетки, как правило, представляют собой эукариотические клетки, такие как клетки млекопитающих, и, как правило, представляют собой клетки человека. В некоторых вариантах осуществления клетки, происходящие из крови, костного мозга, лимфы или лимфоидных органов, представляют собой клетки иммунной системы, такие как клетки врожденного или адаптивного иммунитета, например миелоидные или лимфоидные клетки, включая лимфоциты, обычно Т-клетки и/или NK-клетки. Другие иллюстративные клетки включают стволовые клетки, такие как мультипотентные и плюрипотентные стволовые клетки, в том числе индуцированные плюрипотентные стволовые клетки (iPSC). Клетки обычно представляют собой первичные клетки, такие как клетки, выделенные непосредственно у индивидуума и/или выделенные у индивидуума и замороженные. В некоторых вариантах осуществления клетки включают одно или более подмножеств Т-клеток или других типов клеток, таких как целые популяции Т-клеток, CD4+ клетки, CD8+ клетки и их субпопуляции, такие как те, которые определяются функцией, состоянием активации, зрелостью, потенциалом для дифференцировки, экспансией, рециркуляцией, локализацией и/или персистенцией, антигенспецифичностью, типом антигенного рецептора, присутствием в конкретном органе или компартменте, профилем секреции маркера или цитокина и/или степенью дифференцировки. Применительно к подлежащему лечению пациенту клетки могут быть аллогенными и/или аутологичными. В число методов входят готовые методы. В некоторых аспектах, таких как для готовых технологий, клетки являются плюрипотентными и/или мультипотентными, такими как стволовые клетки, такие как индуцированные плюрипотентные стволовые клетки (iPSC). В некоторых вариантах осуществления способы включают выделение клеток у пациента, приготовление, обработку, культивирование и/или конструирование их, как описано в настоящем документе, и повторное введение их одному и тому же пациенту до или после криоконсервации.
[0496] Среди подтипов и субпопуляций T-клеток и/или CD4+ и/или CD8+ T-клеток присутствуют наивные T (TN) клетки, эффекторные T-клетки (TEFF), T-клетки памяти и их подтипы, такие как стволовые Т-клетки памяти (TSCM), Т-клетки центральной памяти (TCM), Т-клетки эффекторной памяти (TEM) или терминально дифференцированные эффекторные T-клетки памяти, инфильтрирующие опухоль лимфоциты (TIL), незрелые T-клетки, зрелые T-клетки, хелперные T-клетки, цитотоксический T клетки, ассоциированные со слизистой оболочкой инвариантные Т-клетки (MAIT), природные и адаптивные регуляторные Т-клетки (Treg), хелперные Т-клетки, такие как клетки TH1, клетки TH2, клетки TH3, клетки TH17, клетки TH9, клетки TH22, фолликулярные хелперные T-клетки, альфа/бета Т клетки и дельта/гамма Т клетки.
[0497] В некоторых вариантах осуществления клетки являются клетками-натуральными киллерами (NK). В некоторых вариантах осуществления клетки представляют собой моноциты или гранулоциты, например миелоидные клетки, макрофаги, нейтрофилы, дендритные клетки, тучные клетки, эозинофилы и/или базофилы.
[0498] В некоторых вариантах осуществления клетки включают одну или более нуклеиновых кислот, введенных посредством генной инженерии, и тем самым экспрессируют рекомбинантные или генетически сконструированные продукты таких нуклеиновых кислот. В некоторых вариантах осуществления нуклеиновые кислоты являются гетерологичными, то есть обычно не присутствуют в клетке или образце, полученном из клетки, такой как нуклеиновая кислота, полученная из другого организма или клетки, которая, например, обычно не обнаруживается в клетке, которая была сконструирована, и/или в организме, из которого получена такая клетка. В некоторых вариантах осуществления нуклеиновые кислоты не встречаются в природе, такие как нуклеиновая кислота, не встречающаяся в природе, включая такую, которая содержит химерные комбинации нуклеиновых кислот, кодирующих различные домены из множества различных типов клеток.
[0499] В некоторых вариантах осуществления гены и/или генные продукты (и/или их экспрессия) в предложенных клетках и/или композициях, содержащих такие клетки, восстанавливаются, удаляются, устраняются, нокаутируются или нарушаются. Такие гены и/или генные продукты в некоторых аспектах включают один или более генов, кодирующих (или их продукт) константную область TCR альфа (TRAC) и/или константную область TCR бета (TRBC; кодируется у людей TRBC1 или TRBC2), например, чтобы уменьшить или предотвратить экспрессию эндогенного TCR и/или его цепи в клетке, например Т-клетке. В некоторых вариантах осуществления гены и/или генные продукты, такие как TRAC и/или TRBC, уменьшены, удалены, устранены, нокаутированы или нарушены в любой из сконструированных клеток, предложенных в настоящем описании, и/или в любом из способов получения сконструированных клеток, предложенных в настоящем описании. В некоторых вариантах осуществления сконструированные клетки и/или сконструированные клетки, полученные способами, представляют собой клетки, которые были сконструированы для экспрессии связывающей молекулы, описанной в настоящем документе, популяции таких клеток, композиции, содержащие такие клетки и/или обогащенные такими клетками. В некоторых вариантах осуществления гены и/или генные продукты, такие как TRAC и/или TRBC, уменьшены, удалены, устранены, нокаутированы или нарушены в первичных Т-клетках, чтобы уменьшить, удалить, устранить, нокаутировать или нарушить экспрессию эндогенного TCR в первичных Т-клетках, например, сконструированных для экспрессии любого из Т-клеточных рецепторов, описанных в настоящем документе.
[0500] В некоторых вариантах осуществления уменьшение, удаление, устранение, нокаут или нарушение эндогенных генов, кодирующих TCR или цепь, домен и/или его область, осуществляют, например, любыми способами или процессами, описанными в настоящем документе, например в разделе V ниже.
A. Получение клеток для генетического конструирования
[0501] В некоторых вариантах осуществления получение сконструированных клеток включает одну или более стадий культивирования и/или получения. Клетки для введения связывающей молекулы, например TCR или CAR, могут быть выделены из образца, такого как биологический образец, например, полученный у пациента или происходящий от него. В некоторых вариантах осуществления пациент, у которого выделена клетка, является пациентом, имеющим заболевание или состояние или нуждающимся в клеточной терапии, или которому будет назначаться клеточная терапия. Пациентом в некоторых вариантах осуществления является человек, нуждающийся в конкретном терапевтическом вмешательстве, таком как адоптивная клеточная терапия, для которой клетки выделяют, обрабатывают и/или конструируют.
[0502] Соответственно, клетки в некоторых вариантах осуществления являются первичными клетками, например первичными клетками человека. Образцы включают ткани, жидкости и другие образцы, взятые непосредственно у пациента, а также образцы, полученные в результате одной или более стадий обработки, таких как разделение, центрифугирование, генетическое конструирование (например, трансдукция вирусным вектором), промывание и/или инкубация. Биологический образец может быть образцом, полученным непосредственно из биологического источника, или образцом, который обрабатывается. Биологические образцы включают, но не ограничиваются ими, жидкости организма, такие как кровь, плазма, сыворотка, спинномозговая жидкость, синовиальная жидкость, моча и пот, образцы тканей и органов, включая обработанные образцы, полученные из них.
[0503] В некоторых аспектах образец, из которого получены или выделены клетки, представляет собой кровь или образец, полученный из крови, или является или получен из продукта для афереза или лейкафереза. Примеры образцов включают цельную кровь, мононуклеарные клетки периферической крови (РВМС), лейкоциты, костный мозг, тимус, биопсию ткани, опухоль, лейкоз, лимфому, лимфатический узел, лимфоидную ткань, связанную со слизистой оболочкой кишечника, селезенку, другие лимфоидные ткани, печень, легкое, желудок, кишечник, толстую кишку, почки, поджелудочную железу, молочнойую железу, кость, предстательную железу, шейку матки, яички, яичники, миндалины или другой орган и/или клетки, полученные из них. Образцы включают, в контексте клеточной терапии, например адоптивной клеточной терапии, образцы из аутологичных и аллогенных источников.
[0504] В некоторых вариантах осуществления клетки получены из клеточных линий, например, Т-клеточных линий. Клетки в некоторых вариантах осуществления получают из ксеногенного источника, например, от мыши, крысы, примата, не являющегося человеком, или свиньи.
[0505] В некоторых вариантах осуществления выделение клеток включает одну или более стадий получения и/или неаффинного подхода разделения клеток. В некоторых примерах клетки промывают, центрифугируют и/или инкубируют в присутствии одного или более реагентов, например, для удаления нежелательных компонентов, обогащения желаемыми компонентами, лизиса или удаления клеток, чувствительных к конкретным реагентам. В некоторых примерах клетки разделяются на основе одного или более свойств, таких как плотность, адгезивные свойства, размер, чувствительность и/или устойчивость к конкретным компонентам.
[0506] В некоторых примерах клетки из циркулирующей крови пациента получают, например, путем афереза или лейкафереза. Образцы, в некоторых аспектах, содержат лимфоциты, включая Т-клетки, моноциты, гранулоциты, В-клетки, другие ядросодержащие лейкоциты, эритроциты и/или тромбоциты, и в некоторых аспектах содержат клетки, отличные от эритроцитов и тромбоцитов.
[0507] В некоторых вариантах осуществления клетки крови, собранные у пациента, промывают, например, для удаления фракции плазмы и помещения клеток в соответствующий буфер или среду для последующих стадий обработки. В некоторых вариантах осуществления клетки промывают фосфатно-солевым буфером (PBS). В некоторых вариантах осуществления в промывочном растворе отсутствует кальций и/или магний, и/или множество, или всех двухвалентные катионы. В некоторых аспектах стадия промывки выполняется полуавтоматической «проточной» центрифугой (например, клеточным процессором Cobe 2991, Baxter) в соответствии с инструкциями производителя. В некоторых аспектах стадия промывки выполняется посредством фильтрации с тангенциальным потоком (TFF) в соответствии с инструкциями производителя. В некоторых вариантах осуществления клетки ресуспендируют в различных биосовместимых буферах после промывки, таких как, например, PBS без Ca++/Mg++. В некоторых вариантах осуществления компоненты образца клеток крови удаляют, и клетки непосредственно ресуспендируют в культуральной среде.
[0508] В некоторых вариантах осуществления способы включают методы разделения клеток на основе плотности, такие как получение лейкоцитов из периферической крови путем лизиса эритроцитов и центрифугирования в градиенте Перколла или Фиколла.
[0509] В некоторых вариантах осуществления способы выделения включают разделение клеток различных типов на основе экспрессии или наличия в клетке одной или более специфических молекул, таких как поверхностные маркеры, например поверхностные белки, внутриклеточные маркеры или нуклеиновая кислота. В некоторых вариантах осуществления может использоваться любой известный способ разделения на основе таких маркеров. В некоторых вариантах осуществления разделение представляет собой разделение на основе аффинности или иммуноаффинности. Например, выделение в некоторых аспектах включает разделение клеток и клеточных популяций на основе экспрессии клеток или уровня экспрессии одного или более маркеров, обычно маркеров клеточной поверхности, например, путем инкубации с антителом или партнером по связыванию, который специфически связывается с такими маркерами, за которыми обычно следуют стадии промывки и разделения клеток, содержащих связанное антитело или партнер связывания, от клеток, содержащих не связанное антитело или партнер связывания.
[0510] Такие стадии разделения могут быть основаны на положительном отборе, при котором клетки, содержащие связанные реагенты, сохраняются для дальнейшего использования, и/или на отрицательном отборе, при котором клетки, не связанные с антителом или партнером по связыванию, сохраняются. В некоторых примерах обе фракции сохраняются для дальнейшего использования. В некоторых аспектах отрицательный отбор может быть особенно полезным, когда нет доступных антител, которые бы конкретно идентифицировали тип клеток в гетерогенной популяции, так что разделение лучше всего проводить на основе маркеров, экспрессируемых клетками, отличными от желаемой популяции.
[0511] Разделение не должно приводить к 100% обогащению или удалению конкретной клеточной популяции или клеток, экспрессирующих определенный маркер. Например, положительный отбор или обогащение клетками определенного типа, такими как клетки, экспрессирующие маркер, относится к увеличению количества или процента таких клеток, но не обязательно приводит к полному отсутствию клеток, не экспрессирующих маркер. Аналогично, отрицательный отбор, удаление или истощение клеток определенного типа, таких как клетки, экспрессирующие маркер, относится к уменьшению количества или процента таких клеток, но не должно приводить к полному удалению всех таких клеток.
[0512] В некоторых примерах выполняется несколько раундов стадий разделения, где положительно или отрицательно отобранная фракция из одной стадии подвергается другой стадии разделения, такой как последующий положительный или отрицательный отбор. В некоторых примерах одна стадия разделения может истощать клетки, экспрессирующие множество маркеров одновременно, например, путем инкубации клеток с множеством антител или партнеров по связыванию, каждый из которых специфичен для маркера, на которого нацелен отрицательный отбор. Аналогичным образом, несколько типов клеток могут быть одновременно положительно отобраны путем инкубации клеток с множеством антител или партнеров по связыванию, экспрессируемых на различных типах клеток.
[0513] Например, в некоторых аспектах специфические субпопуляции Т-клеток, такие как клетки, положительные или экспрессирующие на высоком уровне один или более поверхностных маркеров, например, CD28 +, CD62L +, CCR7 +, CD27 +, CD127 +, CD4+, CD8+, CD45RA+и/или CD45RO+Т-клетки выделяются с помощью методов положительного или отрицательного отбора.
[0514] Например, CD3 +, CD28+T-клетки могут быть положительно отобраны с использованием анти-CD3/анти-CD28 конъюгированных магнитных гранул (например, DYNABEADS® M-450 CD3/CD28 T Expander Expander).
[0515] В некоторых вариантах осуществления выделение осуществляется путем обогащения определенной популяции клеток путем положительного отбора или истощения определенной популяции клеток путем отрицательного отбора. В некоторых вариантах осуществления положительный или отрицательный отбор осуществляется путем инкубации клеток с одним или более антителами или другими связывающими агентами, которые специфически связываются с одним или более поверхностными маркерами, экспрессируемыми или экспрессируемыми (маркер +) на относительно более высоком уровне (высокий уровень маркера) на положительно или отрицательно отобранных клетках, соответственно.
[0516] В некоторых вариантах осуществления Т-клетки отделяют от образца РВМС путем отрицательного отбора маркеров, экспрессируемых на клетках, отличных от Т-клеток, таких как В-клетки, моноциты или другие лейкоциты, такие как CD14. В некоторых аспектах стадия отбора CD4+ или CD8+ используется для разделения CD4+ хелперных и CD8+ цитотоксических Т-клеток. Такие популяции CD4+ и CD8+ могут быть далее отсортированы в субпопуляции путем положительного или отрицательного отбора по маркерам, экспрессируемым или экспрессируемым в относительно более высокой степени в одной или более субпопуляциях наивных, клеток памяти и/или эффекторных Т-клеток.
[0517] В некоторых вариантах осуществления CD8+ клетки дополнительно обогащаются или истощаются по отношению к наивным клеткам, клеткам центральной памяти, эффекторной памяти и/или стволовым клеткам центральной памяти, как например с помощью положительного или отрицательного отбора на основе поверхностных антигенов, связанных с соответствующей субпопуляцией. В некоторых вариантах осуществления обогащение Т-клетками центральной памяти (TCM) проводят для повышения эффективности, например, для улучшения долгосрочной выживаемости, экспансии и/или трансплантации после введения, которые в некоторых аспектах являются особенно устойчивыми в таких субпопуляциях. Смотри Terakuraet al. (2012) Blood.1:72-82; Wang et al. (2012) J Immunother. 35 (9): 689-701. В некоторых вариантах осуществления объединение TCM-обогащенных CD8+ T-клеток и CD4+ T-клеток дополнительно повышает эффективность.
[0518] В вариантах осуществления Т-клетки памяти присутствуют как в CD62L +, так и в CD62L- субпопуляциях CD8+ лимфоцитов периферической крови. PBMC могут быть обогащены или истощены по фракциям CD62L-CD8+ и/или CD62L+CD8+, например, с использованием антител против CD8 и против CD62L.
[0519] В некоторых вариантах осуществления обогащение Т-клетками центральной памяти (TCM) основано на положительной или высокой поверхностной экспрессии CD45RO, CD62L, CCR7, CD28, CD3 и/или CD 127; в некоторых аспектах оно основано на отрицательном отборе клеток, экспрессирующих или высокоэкспрессирующих CD45RA и/или гранзим B. В некоторых аспектах выделение популяции CD8+, обогащенной клетками TCM, осуществляется путем истощения клеток, экспрессирующих CD4, CD14, CD45RA, и положительного отбора или обогащения клетками, экспрессирующими CD62L. В одном аспекте обогащение Т-клетками центральной памяти (TCM) осуществляют, начиная с отрицательной фракции клеток, отобранных на основе экспрессии CD4, которая подвергается отрицательному отбору на основе экспрессии CD14 и CD45RA и положительному отбору на основе CD62L. Такой отбор в некоторых аспектах выполняется одновременно, а в других аспектах - последовательно, в любом порядке. В некоторых аспектах одна и та же стадия отбора на основе экспрессии CD4, используемая при получении популяции или субпопуляции клеток CD8+, также используется для генерирования популяции или субпопуляции клеток CD4+, так что как положительные, так и отрицательные фракции из разделения на основе CD4 сохраняются и используются на последующих стадиях методов, необязательно после одной или более дополнительных стадий положительного или отрицательного отбора.
[0520] В конкретном примере образец PBMC или другой образец лейкоцитов подвергают селекции клеток CD4+, где сохраняются как отрицательная, так и положительная фракции. Отрицательная фракция затем подвергается отрицательному отбору на основе экспрессии CD14 и CD45RA и положительному отбору на основе маркерной характеристики Т-клеток центральной памяти, такой как CD62L или CCR7, где положительный и отрицательный отборы проводятся в любом порядке.
[0521] CD4+ T-хелперные клетки сортируются на наивные, клетки центральной памяти и эффекторные клетки путем идентификации популяций клеток, которые имеют антигены клеточной поверхности. CD4+ лимфоциты могут быть получены стандартными методами. В некоторых вариантах осуществления наивными CD4+ T-лимфоцитами являются CD45RO-, CD45RA +, CD62L +, CD4+ T-клетки. В некоторых вариантах осуществления CD4+ клетки центральной памяти представляют собой CD62L+и CD45RO +. В некоторых вариантах осуществления эффекторные CD4+ клетки представляют собой CD62L- и CD45RO-.
[0522] В одном примере для обогащения клеток CD4+ путем отрицательного отбора коктейль из моноклональных антител обычно включает антитела к CD14, CD20, CD11b, CD16, HLA-DR и CD8. В некоторых вариантах осуществления антитело или связывающий партнер связаны с твердой подложкой или матрицей, такой как магнитная гранула или парамагнитная гранула, чтобы обеспечить разделение клеток для положительного и/или отрицательного отбора. Например, в некоторых вариантах осуществления клетки и популяции клеток разделяют или выделяют с использованием иммуномагнитных (или аффинно-магнитных) методов разделения (рассмотрено в Methods in Molecular Medicine, vol. 58: Metastasis Research Protocols, Vol. 2: Cell Behavior In Vitro and In Vivo, p 17-25 Edited by: S. A. Brooks and U. Schumacher © Humana Press Inc., Totowa, NJ).
[0523] В некоторых аспектах образец или композицию клеток, подлежащих разделению, инкубируют с небольшим намагничиваемым или магнитно-чувствительным материалом, таким как магнитно-чувствительные частицы или микрочастицы, такие как парамагнитные гранулы (например, такие как гранулы Dynabeads или MACS). Магнитно-чувствительный материал, например частица, обычно прямо или косвенно связан с партнером по связыванию, например антителом, которое специфически связывается с молекулой, например поверхностным маркером, присутствующим на клетке, клетках или популяции клеток, которые желательно отделить, например, то, что желательно подвергнуть отрицательному или положительному отбору.
[0524] В некоторых вариантах осуществления магнитная частица или гранула содержат магнитно-чувствительный материал, связанный с конкретным связывающим элементом, таким как антитело или другой связывающий партнер. Существует много хорошо известных магнитно-чувствительных материалов, используемых в методах магнитного разделения. Подходящие магнитные частицы включают частицы, описанные у Molday, Патенты США 4452773 и в Европейском патенте EP 452342 B, которые включены в настоящее описание в качестве ссылки. Частицы коллоидного размера, такие как описанные у Owen, Патенты США 4795698 и у Liberti et al., Патенты США № 5200084 являются другими примерами.
[0525] Инкубацию обычно проводят в условиях, при которых антитела или партнеры по связыванию или молекулы, такие как вторичные антитела или другие реагенты, которые специфически связываются с такими антителами или партнерами по связыванию, которые прикреплены к магнитной частице или грануле, специфически связываются с молекулами клеточной поверхности, если они присутствуют на клетках в образце.
[0526] В некоторых аспектах образец помещают в магнитное поле, и те клетки, к которым прикреплены магниточувствительные или намагничиваемые частицы, будут притягиваться к магниту и отделяться от немеченых клеток. Для положительного отбора клетки, которые притягиваются к магниту, сохраняются; для отрицательного отбора сохраняются клетки, которые не притягиваются (клетки без метки). В некоторых аспектах комбинация положительного и отрицательного отбора выполняется во время одной и той же стадии отбора, где положительные и отрицательные фракции сохраняются и дополнительно обрабатываются или подвергаются дополнительным стадиям разделения.
[0527] В некоторых вариантах осуществления магнитно-чувствительные частицы покрыты первичными антителами или другими партнерами по связыванию, вторичными антителами, лектинами, ферментами или стрептавидином. В некоторых вариантах осуществления магнитные частицы прикрепляются к клеткам посредством покрытия первичными антителами, специфичными для одного или более маркеров. В некоторых вариантах осуществления клетки, а не гранулы, метят первичным антителом или партнером связывания, а затем добавляют магнитные частицы, покрытые специфическим для типа клеток вторичным антителом или другим партнером связывания (например, стрептавидином). В некоторых вариантах осуществления магнитные частицы, покрытые стрептавидином, используются в сочетании с биотинилированными первичными или вторичными антителами.
[0528] В некоторых вариантах осуществления магниточувствительные частицы оставляют прикрепленными к клеткам, которые затем следует инкубировать, культивировать и/или использовать в конструировании; в некоторых аспектах частицы остаются прикрепленными к клеткам для введения пациенту. В некоторых вариантах осуществления намагничиваемые или магниточувствительные частицы удаляются из клеток. Способы удаления намагничиваемых частиц из клеток известны и включают, например, использование конкурирующих немеченых антител, намагничиваемых частиц или антител, конъюгированных с расщепляемыми линкерами, и т. д. В некоторых вариантах осуществления намагничиваемые частицы являются биоразлагаемыми.
[0529] В некоторых вариантах осуществления отбор на основе аффинности осуществляется посредством магнитно-активированной сортировки клеток (MACS) (Miltenyi Biotec, Оберн, Калифорния). Системы магнитно-активированной сортировки клеток (MACS) способны отбирать клетки высокой чистоты с намагниченными частицами, прикрепленными к ним. В некоторых вариантах осуществления MACS работает в режиме, в котором нецелевые и целевые образцы последовательно элюируются после приложения внешнего магнитного поля. То есть клетки, прикрепленные к намагниченным частицам, удерживаются на месте, в то время как незакрепленные частицы элюируются. Затем, после того, как этот первая стадия элюирования завершена, образцы, которые были пойманы в ловушку в магнитном поле и не были элюированы, освобождаются каким-либо образом, так что их можно элюировать и восстанавливать. В некоторых вариантах осуществления нецелевые клетки метят и истощают из гетерогенной популяции клеток.
[0530] В некоторых вариантах осуществления выделение или разделение осуществляют с использованием системы, устройства или аппарата, которые выполняют одну или более стадий способов из стадий выделения, подготовки, разделения, обработки, инкубации, культивирования и/или стадий приготовления. В некоторых аспектах система используется для выполнения каждой из этих стадий в закрытой или стерильной среде, например, чтобы минимизировать ошибки, манипуляции пользователя и/или загрязнение. В одном примере система представляет собой систему, как описано в международной заявке на патент, номер публикации WO2009/072003 или US 2011/0003380 A1.
[0531] В некоторых вариантах осуществления система или аппарат выполняют один или более, например, все стадии выделения, обработки, конструирования и приготовления состава в интегрированной или автономной системе и/или автоматизированным или программируемым образом. В некоторых аспектах система или аппарат включают в себя компьютер и/или компьютерную программу, связанную с системой или аппаратом, которые позволяют пользователю программировать, контролировать, оценивать результат и/или регулировать различные аспекты стадий обработки, выделения, конструирования и приготовления состава.
[0532] В некоторых аспектах разделение и/или другие стадии проводят с использованием системы CliniMACS (Miltenyi Biotec), например, для автоматического разделения клеток на уровне клинического масштаба в закрытой и стерильной системе. Компоненты могут включать в себя встроенный микрокомпьютер, блок магнитного разделения, перистальтическую помпу и различные пережимные клапаны. Встроенный компьютер в некоторых аспектах контролирует все компоненты прибора и дает указание системе выполнять повторяющиеся процедуры в стандартизированной последовательности. Блок магнитного разделения в некоторых аспектах включает в себя подвижный постоянный магнит и держатель для селекционной колонки. Перистальтическая помпа контролирует скорость потока по всем трубкам и вместе с пережимными клапанами обеспечивает контролируемый поток буфера через систему и постоянную суспензию клеток.
[0533] Система CliniMACS в некоторых аспектах использует связанные с антителами намагничиваемые частицы, которые поставляются в стерильном непирогенном растворе. В некоторых вариантах осуществления после мечения клеток магнитными частицами клетки промывают для удаления избытка частиц. Затем пакет с препаратом клеток соединяется с комплектом трубок, которые, в свою очередь, соединяются с пакетом, содержащим буфер, и с пакетом для сбора клеток. Комплект трубок состоит из предварительно собранных стерильных трубок, включая предварительную колонку и разделительную колонку, и предназначен только для одноразового использования. После запуска программы разделения система автоматически наносит образец клеток на разделительную колонку. Меченые клетки задерживаются в колонке, в то время как немеченые клетки удаляются с помощью серии стадий промывки. В некоторых вариантах осуществления клеточные популяции для использования со способами, описанными в настоящем документе, являются немечеными и не задерживаются в колонке. В некоторых вариантах осуществления клеточные популяции для использования со способами, описанными в настоящем документе, помечены и задерживаются в колонке. В некоторых вариантах осуществления клеточные популяции для использования в описанных в настоящем документе способах элюируют из колонки после удаления магнитного поля и собирают в пакет для сбора клеток.
[0534] В некоторых вариантах осуществления разделение и/или другие стадии проводят с использованием системы CliniMACS Prodigy (Miltenyi Biotec). Система CliniMACS Prodigy в некоторых аспектах оснащена блоком обработки клеток, который позволяет автоматически промывать и фракционировать клетки центрифугированием. Система CliniMACS Prodigy может также включать в себя встроенную камеру и программное обеспечение для распознавания изображений, которое определяет оптимальную конечную точку фракционирования клеток путем определения макроскопических слоев исходного клеточного продукта. Например, периферическая кровь может быть автоматически разделена на эритроциты, лейкоциты и слои плазмы. Система CliniMACS Prodigy также может включать в себя интегрированную камеру для культивирования клеток, которая выполняет протоколы культивирования клеток, такие как, например, дифференцировка и экспансия клеток, загрузка антигена и продолжительное культивирование клеток. Входные порты могут обеспечить стерильное удаление и пополнение среды, и клетки можно контролировать с помощью встроенного микроскопа. Смотри, например, Klebanoff et al. (2012) J Immunother. 35 (9): 651-660, Terakuraet al. (2012) Blood.1: 72-82, и Wang et al. (2012) J Immunother. 35 (9): 689-701.
[0535] В некоторых вариантах осуществления описанную в настоящем документе популяцию клеток собирают и обогащают (или истощают) с помощью проточной цитометрии, в которой клетки, окрашенные по множеству маркеров клеточной поверхности, переносятся в жидком потоке. В некоторых вариантах осуществления описанную в настоящем документе популяцию клеток собирают и обогащают (или истощают) посредством сортировки в препаративном масштабе (FACS). В некоторых вариантах осуществления популяцию клеток, описанную в настоящем документе, собирают и обогащают (или истощают) путем использования чипов микроэлектромеханических систем (MEMS) в комбинации с системой детектирования на основе FACS (см., например, WO 2010/033140, Cho et al. (2010) Lab Chip 10, 1567-1573; и Godin et al. (2008) J Biophoton. 1 (5): 355-376. В обоих случаях клетки могут быть помечены несколькими маркерами, что позволяет выделить четко определенные субпопуляции Т-клеток с высокой степенью чистоты.
[0536] В некоторых вариантах осуществления антитела или партнеры связывания помечены одним или более детектируемыми маркерами для облегчения разделения для положительного и/или отрицательного отбора. Например, разделение может быть основано на связывании с флуоресцентно меченными антителами. В некоторых примерах разделение клеток на основе связывания антител или других партнеров связывания, специфичных для одного или более маркеров клеточной поверхности, осуществляется в жидком потоке, таком как сортировка активированных флуоресценцией клеток (FACS), включая препаративный масштаб (FACS) и/или чипы микроэлектромеханических систем (MEMS), например, в комбинации с проточной цитометрической системой детектирования. Такие методы допускают положительный и отрицательный отбор на основе множества маркеров одновременно.
[0537] В некоторых вариантах осуществления способы получения включают стадии замораживания, например, криоконсервации клеток, до или после выделения, инкубации и/или конструирования. В некоторых вариантах осуществления стадия замораживания и последующего оттаивания удаляет гранулоциты и, в некоторой степени, моноциты в клеточной популяции. В некоторых вариантах осуществления клетки суспендируют в замораживающем растворе, например, после стадии промывания для удаления плазмы и тромбоцитов. В некоторых аспектах может использоваться любое из множества известных решений и параметров замораживания. Один пример включает использование PBS, содержащего 20% DMSO и 8% человеческого сывороточного альбумина (HSA), или другие подходящие среды для замораживания клеток. Затем его разбавляют 1:1 средой, так что конечная концентрация DMSO и HSA составляет 10% и 4%, соответственно. Затем клетки замораживают до -80°С со скоростью 1°С в минуту и хранят в паровой фазе резервуара для хранения жидкого азота.
[0538] В некоторых вариантах осуществления предложенные способы включают стадии культивирования, инкубации, культивации и/или генетического конструирования. Например, в некоторых вариантах осуществления предложены способы инкубации и/или конструирования истощенных клеточных популяций и композиций, инициирующих культивирование.
[0539] Таким образом, в некоторых вариантах осуществления клеточные популяции инкубируют в инициирующей культуру композиции. Инкубацию и/или конструирование можно проводить в культуральном сосуде, таком как блок, камера, лунка, колонка, пробирка, набор трубок, клапан, флакон, культуральная чашка, пакет или другой контейнер для культивирования или культивации клеток.
[0540] В некоторых вариантах осуществления клетки инкубируют и/или культивируют до или в связи с генетическим конструированием. Стадии инкубации могут включать культивирование, культивацию, стимуляцию, активацию и/или размножение. В некоторых вариантах осуществления композиции или клетки инкубируют в присутствии стимулирующих условий или стимулирующего агента. Такие условия включают условия, предназначенные для того, чтобы индуцировать пролиферацию, экспансию, активацию и/или выживание клеток в популяции, имитировать воздействие антигена и/или праймировать клетки для генетического конструирования, как например для введения антигенного рецептора.
[0541] Условия могут включать одно или более из конкретных сред, температуры, содержания кислорода, содержания диоксида углерода, времени, агентов, например, питательных веществ, аминокислот, антибиотиков, ионов и/или стимулирующих факторов, таких как цитокины, хемокины, антигены, связывание партнеры, слитые белки, рекомбинантные растворимые рецепторы и любые другие агенты, предназначенные для активации клеток.
[0542] В некоторых вариантах осуществления стимулирующие условия или агенты включают один или более агентов, например лиганд, который способен активировать внутриклеточный сигнальный домен комплекса TCR. В некоторых аспектах агент включает или инициирует внутриклеточный сигнальный каскад TCR/CD3 в Т-клетке. Такие агенты могут включать антитела, такие как антитела, специфичные для компонента TCR и/или костимулирующего рецептора, например, антитело против CD3. В некоторых вариантах осуществления стимулирующие условия включают один или более агентов, например, лиганд, который способен стимулировать костимулирующий рецептор, например антитело против CD28. В некоторых вариантах осуществления такие агенты и/или лиганды могут быть связаны с твердой подложкой, такой как гранула, и/или одним или более цитокинами. Необязательно, способ экспансии может дополнительно включать стадию добавления антитела против CD3 и/или антитела против CD28 к культуральной среде (например, в концентрации, по меньшей мере, примерно 0,5 нг/мл). В некоторых вариантах осуществления стимулирующие агенты включают IL-2, IL-15 и/или IL-7. В некоторых аспектах концентрация IL-2 составляет, по меньшей мере, примерно 10 единиц/мл.
[0543] В некоторых аспектах инкубацию проводят в соответствии с методиками, такими как описанные в патенте США № 6040177, выданном Riddell et al., Klebanoff et al. (2012) J. Immunother. 35 (9): 651-660, Terakuraet al. (2012) Blood.1: 72-82 и/или Wang et al. (2012) J Immunother. 35 (9): 689-701.
[0544] В некоторых вариантах осуществления Т-клетки наращивают путем добавления композиции фидерных клеток, инициирующей культуру, таких как неделящиеся мононуклеарные клетки периферической крови (РВМС) (например, так, чтобы полученная популяция клеток содержала, по меньшей мере, примерно 5, 10, 20 или 40 или более фидерных клеток РВМС для каждого Т-лимфоцита в исходной популяции, которая подлежит наращиванию); и путем инкубации культуры (например, в течение времени, достаточного для наращивания количества Т-клеток). В некоторых аспектах неделящиеся фидерные клетки могут содержать гамма-облученные фидерные клетки РВМС. В некоторых вариантах осуществления РВМС облучают гамма-излучением в диапазоне от примерно 3000 до 3600 рад для предотвращения деления клеток. В некоторых аспектах фидерные клетки добавляют в культуральную среду до добавления популяций Т-клеток.
[0545] В некоторых вариантах осуществления стимулирующие условия включают температуру, подходящую для роста человеческих Т-лимфоцитов, например, по меньшей мере, примерно 25 градусов Цельсия, обычно, по меньшей мере, примерно 30 градусов и, как правило или примерно 37 градусов Цельсия. Необязательно, инкубация может дополнительно включать добавление неделящихся EBV-трансформированных лимфобластоидных клеток (LCL) в качестве фидерных клеток. LCL можно облучать гамма-излучением в диапазоне от 6000 до 10000 рад. Фидерные клетки LCL в некоторых аспектах представлены в любом подходящем количестве, таком как отношение фидерных клеток LCL к исходным Т-лимфоцитам, по меньшей мере, примерно 10:1.
[0546] В вариантах осуществления антиген-специфические Т-клетки, такие как антиген-специфические CD4+ и/или CD8+ Т-клетки, получают путем стимуляции наивных или антиген-специфических Т-лимфоцитов антигеном. Например, антигенспецифические линии или клоны Т-клеток могут генерироваться для антигенов цитомегаловируса путем выделения Т-клеток от инфицированных пациентов и путем стимуляции клеток in vitro тем же антигеном.
B. Векторы и методы для генетического конструирования
[0547] Также предложены способы, нуклеиновые кислоты, композиции и наборы для экспрессии связывающих молекул и для получения генетически сконструированных клеток, экспрессирующих такие связывающие молекулы. Генетическое конструирование обычно включает введение нуклеиновой кислоты, кодирующей связывающую молекулу, например, TCR или CAR, например TCR-подобного CAR, в клетку, как например путем ретровирусной трансдукции, трансфекции или трансформации.
[0548] В некоторых вариантах осуществления перенос гена осуществляется сначала путем стимуляции клетки, например, путем объединения ее со стимулом, который индуцирует ответ, такой как пролиферация, выживание и/или активация, например, как измеряется экспрессией цитокина или маркера активации, с последующей трансдукцией активированных клеток и экспансией в культуре до количества, достаточного для клинического применения.
[0549] В некоторых случаях избыточная экспрессия стимулирующего фактора (например, лимфокина или цитокина) может быть токсичной для пациента. Таким образом, в некоторых контекстах сконструированные клетки включают генные сегменты, которые вызывают восприимчивость клеток к отрицательному отбору in vivo, например, при введении в адоптивной иммунотерапии. Например, в некоторых аспектах клетки сконструированы таким образом, что они могут быть удалены в результате изменения состояния in vivo пациента, которому их вводят. Отрицательный селектируемый фенотип может быть результатом включения гена, который придает чувствительность к вводимому агенту, например, соединению. Отрицательные селектируемые гены включают ген тимидинкиназы вируса простого герпеса типа I (HSV-I TK) (Wigler et al., Cell 2: 223, 1977), который придает чувствительность к ганцикловиру; ген клеточной гипоксантинфосфрибозилтрансферазы (HPRT), ген клеточной аденинфосфорибозилтрансферазы (APRT), бактериальная цитозиндеаминаза (Mullen et al., Proc. Natl. Acad. Sci. USA. 89:33 (1992)).
[0550] В некоторых аспектах клетки дополнительно сконструированы для стимулирования экспрессии цитокинов или других факторов. Различные способы введения генетически сконструированных компонентов хорошо известны и могут использоваться с предложенными способами и композициями. Примеры способов включают способы переноса нуклеиновых кислот, кодирующих связывающие молекулы, в том числе с помощью вируса, например ретровируса или лентивируса, трансдукции, транспозонов и электропорации.
[0551] В некоторых вариантах осуществления рекомбинантные нуклеиновые кислоты переносятся в клетки с использованием рекомбинантных инфекционных вирусных частиц, таких как, например, векторы, полученные из обезьяньего вируса 40 (SV40), аденовирусов, аденоассоциированного вируса (AAV). В некоторых вариантах осуществления рекомбинантные нуклеиновые кислоты переносятся в Т-клетки с использованием рекомбинантных лентивирусных векторов или ретровирусных векторов, таких как гамма-ретровирусные векторы (см., например, Koste et al. (2014) Gene Therapy 2014 Apr 3. doi: 10.1038/gt.2014.25; Carlens et al. (2000) Exp Hematol 28(10): 1137-46; Alonso-Camino et al. (2013) Mol Ther Nucl Acids 2, e93; Park et al., Trends Biotechnol. 2011 November 29(11): 550-557.
[0552] В некоторых вариантах осуществления ретровирусный вектор имеет последовательность длинного концевого повтора (LTR), например ретровирусный вектор, полученный из вируса мышиного лейкоза Молони (MoMLV), вируса миелопролиферативной саркомы (MPSV), вируса мышиных эмбриональных стволовых клеток (MESV), мышиного вируса стволовых клеток (MSCV), вируса некроза селезенки (SFFV). Большинство ретровирусных векторов происходят из мышиных ретровирусов. В некоторых вариантах осуществления ретровирусы включают те, которые получены из любого источника клеток птиц или млекопитающих. Ретровирусы, как правило, являются амфотропными, что означает, что они способны инфицировать клетки-хозяева нескольких видов, включая человека. В одном варианте осуществления ген, подлежащий экспрессии, заменяет ретровирусные последовательности gag, pol и/или env. Был описан ряд иллюстративных ретровирусных систем (например, Патенты США No. 5219740; 6207453; 5219740; Miller and Rosman (1989) BioTechniques 7:980-990; Miller, A. D. (1990) Human Gene Therapy 1:5-14; Scarpa et al. (1991) Virology 180:849-852; Burns et al. (1993) Proc. Natl. Acad. Sci. USA 90:8033-8037; и Boris-Lawrie and Temin (1993) Cur. Opin. Genet. Develop. 3: 102-109.
[0553] Методы лентивирусной трансдукции известны. Иллюстративные способы описаны, например, в Wang et al. (2012) J. Immunother. 35(9): 689-701; Cooper et al. (2003) Blood. 101:1637-1644; Verhoeyen et al. (2009) Methods Mol Biol. 506: 97-114; и Cavalieri et al. (2003) Blood. 102(2): 497-505.
[0554] В некоторых вариантах осуществления рекомбинантные нуклеиновые кислоты переносятся в Т-клетки посредством электропорации (см., например, Chicaybam et al., (2013) PLoS ONE8 (3): e60298 и Van Tedeloo et al. (2000) Gene Therapy 7 (16): 1431-1437). В некоторых вариантах осуществления рекомбинантные нуклеиновые кислоты переносятся в Т-клетки посредством транспозиции (см., например, Manuri et al. (2010) Hum Gene Ther 21(4): 427-437; Sharma et al. (2013) Molec Ther Nucl Acids 2, e74; и Huang et al. (2009) Methods Mol Biol 506: 115-126). Другие способы введения и экспрессии генетического материала в иммунных клетках включают трансфекцию с фосфатом кальция (например, как описано в Current Protocols in Molecular Biology, John Wiley & Sons, New York. N.Y.), слияние протопластов, катионную липосомопосредованную трансфекцию; бомбардировку микрочастицами частиц вольфрама (Johnston, Nature, 346: 776-777 (1990)); и копрециапитация ДНК с фосфатом стронция (Brash et al., Mol. Cell Biol., 7: 2031-2034 (1987)).
[0555] Другими подходами и векторами для переноса нуклеиновых кислот, кодирующих связывающие молекулы или рекомбинантные продукты, являются те, которые описаны, например, в международной заявке на патент, публикация №: WO2014/055668 и в Патенте США № 7446190.
[0556] Среди дополнительных нуклеиновых кислот, например, гены для введения - это гены, которые улучшают эффективность терапии, например, путем повышения жизнеспособности и/или функции перенесенных клеток; гены, обеспечивающие генетический маркер для отбора и/или оценки клеток, например, для оценки выживаемости или локализации in vivo; гены для повышения безопасности, например, делая клетку восприимчивой к отрицательному отбору in vivo, как описано в Lupton S. D. et al., Mol. и Cell Biol., 11: 6 (1991); and Riddell et al. Human Human Therapy 3: 319-338 (1992); см. также публикации PCT/US91/08442 и PCT/US94/05601 Lupton et al., описывающие использования бифункциональных селектируемых слитых генов, полученных в результате слияния доминантного положительного селектируемого маркера с отрицательным селектируемым маркером. См., например, Riddell et al., Патент США № 6040177, в столбцах 14-17.
[0557] Таким образом, в некоторых вариантах осуществления предложены сконструированные клетки, такие как клетки, содержащие связывающую молекулу (такую как TCR или его антигенсвязывающий фрагмент, или антитело или его антигенсвязывающий фрагмент), нуклеиновую кислоту или вектор, как описано в настоящем документе. В некоторых аспектах клетку получают путем трансформации клетки in vitro или ex vivo вектором, описанным в настоящем документе. В некоторых аспектах клетка представляет собой Т-клетку, такую как CD8+ или CD4+ Т-клетка. В некоторых вариантах осуществления связывающая молекула является гетерологичной для клетки.
[0558] В некоторых случаях сконструированная клетка содержит гетерологичный TCR или его антигенсвязывающий фрагмент, который распознает или связывается с пептидным эпитопом, полученным из E6 HPV16. В некоторых случаях TCR или его антигенсвязывающий фрагмент не распознает или не связывается с эпитопом E6 (29-38), содержащим аминокислотную последовательность TIHDIILECV (SEQ ID NO: 233). В некоторых случаях TCR или его антигенсвязывающий фрагмент, который распознает или связывается с пептидным эпитопом, полученным из E6 HPV16, представляет собой или содержит последовательность, представленную в SEQ ID NO: 232 или SEQ ID NO: 234.
[0559] В некоторых вариантах осуществления сконструированная клетка содержит гетерологичный TCR или его антигенсвязывающий фрагмент, который распознает или связывается с пептидным эпитопом, полученным из E7 HPV 16. В некоторых вариантах осуществления TCR или его антигенсвязывающий фрагмент не распознает или не связывается с эпитопом E7 (11-19), содержащим аминокислотную последовательность YMLDLQPET (SEQ ID NO: 236). В некоторых случаях TCR или его антигенсвязывающий фрагмент, который распознает или связывается с пептидным эпитопом, полученным из E7 HPV16, представляет собой или содержит последовательность, представленную в любой из SEQ ID NO: 235-239. В некоторых случаях пептид, полученный из E7 HPV16, представляет собой или содержит последовательность, представленную в SEQ ID NO: 235.
V. СПОСОБЫ РЕДАКТИРОВАНИЯ ЭНДОГЕННЫХ ГЕНОВ, КОДИРУЮЩИХ T-КЛЕТОЧНЫЙ РЕЦЕПТОР (TCR), И КОНСТРУИРОВАНИЕ КЛЕТОК ДЛЯ ЭКСПРЕССИИ СВЯЗЫВАЮЩИХ МОЛЕКУЛ ПУТЕМ НАПРАВЛЕННОЙ ИНТЕГРАЦИИ
[0560] В некоторых аспектах предложенные связывающие молекулы, например рекомбинантный T-клеточный рецептор (TCR) или его фрагмент или цепь, экспрессируются в сконструированной клетке, например, сконструированной T-клетке. В некоторых вариантах осуществления предложены генетически сконструированные Т-клетки, экспрессирующие любую из описанных связывающих молекул, например рекомбинантные TCR или их фрагмент или цепь, предложенные в настоящем документе, например, для адоптивной клеточной терапии, и соответствующие композиции, способы, применения и наборы и изделия производства, используемые для осуществления способов. В некоторых аспектах один или более эндогенных генов и/или генных продуктов (и/или их экспрессия) в сконструированных клетках модифицируются, например, путем редактирования генов. В некоторых аспектах редактирование генов приводит к уменьшению, удалению, устранению, нокауту или нарушению одного или более эндогенных генных продуктов (и/или их экспрессии) и/или направленной интеграции экзогенных, гетерологичных или трансгенных последовательностей, например последовательностей, кодирующих связывающую молекулу, например, рекомбинантный TCR, с помощью таких методов, как направляемая гомологией репарация (HDR). В некоторых вариантах осуществления иммунные клетки сконструированы для экспрессии любой из связывающих молекул, например, рекомбинантных TCR, и последовательности, кодирующие связывающую молекулу, например, рекомбинантный TCR, могут быть нацелены на конкретный локус с помощью методов редактирования генов, таких как HDR.
[0561] В некоторых вариантах осуществления один или более эндогенных генов и/или генных продуктов (и/или их экспрессия) в предложенных клетках и/или композициях, содержащих такие клетки, уменьшают, удаляют, устраняют, нокаутируют или нарушают, например, путем введение генетического нарушения, такого как разрыв ДНК. В некоторых вариантах осуществления гены и/или генные продукты уменьшают, удаляют, устраняют, нокаутируют или нарушают в любой из клеток, сконструированных для экспрессии любой из связывающих молекул, например рекомбинантных TCR, предложенных в настоящем описании. В некоторых вариантах осуществления предложены клетки, которые были сконструированы для экспрессии связывающей молекулы, например, рекомбинантного TCR, описанного в настоящем документе, популяции таких клеток, композиции, содержащие такие клетки и/или обогащенные такими клетками.
[0562] В некоторых вариантах осуществления один или более эндогенных генов и/или генных продуктов (и/или их экспрессия) в предложенных клетках и/или композициях, содержащих такие клетки, уменьшают, удаляют, устраняют, нокаутируют или нарушают, включая один или более генов, кодирующих (или их продукты) константную область TCR альфа (TRAC) и/или константную область TCR бета (TRBC; кодируется у человека TRBC1 или TRBC2), например, для уменьшения или предотвращения экспрессии эндогенного TCR и/или его цепи в клетке например, Т-клетке. В некоторых вариантах осуществления гены и/или генные продукты, такие как TRAC и/или TRBC, уменьшены, удалены, утсранены, нокаутированы или нарушены в любой из сконструированных клеток, предложенных в настоящем описании, и/или в любом из способов получения сконструированных клеток, предложенных в настоящем описании. В некоторых вариантах осуществления сконструированные клетки и/или сконструированные клетки, полученные способами, представляют собой клетки, которые были сконструированы для экспрессии связывающей молекулы, описанной в настоящем документе, популяции таких клеток, композиции, содержащие такие клетки и/или обогащенные такими клетками. В некоторых вариантах осуществления гены и/или генные продукты, такие как TRAC и/или TRBC, уменьшены, удалены, устранены, нокаутированы или нарушены в первичных Т-клетках, чтобы уменьшить, удалить, устранить, нокаутировать или нарушить экспрессию эндогенного TCR в первичных Т-клетках, например, сконструированных для экспрессии любого из Т-клеточных рецепторов, описанных в настоящем документе.
[0563] В некоторых вариантах осуществления редактирование генов, например, с помощью HDR, включает: i) введение в иммунную клетку одного или более агентов, способных индуцировать генетическое нарушение одного или более сайтов-мишеней в гене, кодирующем домен, или область альфа-цепи (TCRα) Т-клеточного рецептора и/или один или более генов, кодирующих домен или область бета-цепи Т-клеточного рецептора (TCRβ); и ii) введение в иммунную клетку полинуклеотида, например матричного полинуклеотида, содержащего трансген, кодирующий связывающую молекулу, например рекомбинантный TCR или его цепь, такой как любой из предложенных рекомбинантных TCR, где трансген, кодирующий связывающую молекулу, например рекомбинантный TCR или его цепь, нацелен на один или примерно один из по меньшей мере одного сайта-мишени с помощью направляемой гомологией репарации (HDR).
[0564] В некоторых аспектах полинуклеотиды, например матричные полинуклеотиды, содержащие трансгенную последовательность (также называемую в настоящем документе экзогенной или гетерологичной последовательностью нуклеиновой кислоты), кодирующую связывающую молекулу, например рекомбинантный TCR или его цепь, и последовательности, гомологичные области генетического нарушения, могут быть введены в клетку, содержащую генетическое нарушение в эндогенных локусах TRAC и/или TRBC. В некоторых аспектах при наличии целевого генетического нарушения, например, разрыва ДНК, последовательности нуклеиновой кислоты могут использоваться в качестве матрицы для репарации ДНК, чтобы эффективно копировать и интегрировать последовательность трансгена, например последовательности нуклеиновой кислоты, кодирующие связывающую молекулу, например, рекомбинантный TCR или его цепь, в сайт или примерно в сайт целевого генетического нарушения с помощью HDR, на основе гомологии между последовательностью эндогенного гена, окружающей сайт-мишень, и 5'- и/или 3'-плечами гомологии, включенными в матричный полинуклеотид.
[0565] В некоторых вариантах осуществления генетически сконструированные клетки модифицированы так, чтобы они содержали локус TRAC и/или TRBC, который содержит последовательности нуклеиновой кислоты, кодирующие любую из предложенных связывающих молекул, например, рекомбинантный TCR или его фрагмент. В некоторых аспектах локус TRAC и/или TRBC в генетически сконструированной клетке модифицируется, например, путем редактирования генов, чтобы включать последовательность трансгена, кодирующую связывающую молекулу, например, рекомбинантный TCR или его цепь, которые интегрированы в эндогенный локус TRAC и/или TRBC, который обычно кодирует константные домены TCRα или TCRβ. В некоторых вариантах осуществления редактирование генов включает в себя индукцию целевого генетического нарушения в одном или более эндогенных генах, кодирующих константные домены TCRα или TCRβ, и направляемую гомологичной репарацией (HDR) с использованием одного или более матричных полинуклеотидов, содержащих трансген, кодирующий связывающую молекулу, например, рекомбинантный TCR или его цепь, таким образом направляя интеграцию трансгена в локус TRAC и/или TRBC. В некоторых вариантах осуществления трансген кодирует часть рекомбинантного TCR и интегрируется в рамке в открытую рамку считывания TCR и/или в локус гена. В некоторых вариантах осуществления трансген кодирует часть рекомбинантного TCR и встраивается в рамке внутрь эндогенной открытой рамки считывания, кодирующей константный домен TCR. В некоторых вариантах осуществления интеграция трансгена в локус модифицирует и/или приводит к получению модифицированного локуса, который кодирует полный рекомбинантный TCR.
A. Генетическое нарушение эндогенных генов, кодирующих TCR
[0566] В некоторых вариантах осуществления направленное генетическое нарушение происходит в эндогенных генах, которые кодируют один или более доменов, областей и/или цепей эндогенного Т-клеточного рецептора (TCR). В некоторых вариантах осуществления генетическое нарушение является мишенью в эндогенных генных локусах, которые кодируют TCRα и/или TCRβ. В некоторых вариантах осуществления мишенью генетического нарушения является ген, кодирующий константный домен TCRα (TRAC у людей) и/или константный домен TCRβ (TRBC1 или TRBC2 у человека).
[0567] В некоторых вариантах осуществления гены и/или генные продукты, являющиеся мишенью для уменьшения, делеции, исключения, нокаута или нарушения, представляют собой эндогенные гены, кодирующие TCR или цепь, домен и/или его область. В некоторых вариантах осуществления сайт-мишень для нарушения находится в гене константной области Т-клеточного рецептора альфа (TRAC). В некоторых вариантах осуществления сайт-мишень для нарушения находится в гене константной области 1 Т-клеточного рецептора бета (TRBC1) или константной области 2 Т-клеточного рецептора бета (TRBC2). В некоторых вариантах осуществления один или более сайтов-мишеней находятся в гене TRAC и в одном или в обоих из генов TRBC1 и TRBC2.
[0568] В некоторых вариантах осуществления эндогенный TCR Cα кодируется геном TRAC (номенклатура IMGT). Иллюстративная нуклеотидная последовательность локуса гена константной области Т-клеточного рецептора альфа человека (TRAC) представлена в SEQ ID NO: 348 (Эталонная последовательность NCBI: NG_001332.3, TRAC). В некоторых вариантах осуществления эндогенный TCR Cβ кодируется генами TRBC1 или TRBC2 (номенклатура IMGT). Иллюстративная нуклеотидная последовательность локуса гена константной области 1 Т-клеточного рецептора бета человека (TRBC1) представлена в SEQ ID NO: 349 (Эталонная последовательность NCBI: NG_001333.2, TRBC1); и иллюстративная нуклеотидная последовательность локуса гена константной области 2 Т-клеточного рецептора бета человека (TRBC2) представлена в SEQ ID NO: 1047 (Эталонная последовательность NCBI: NG_001333.2, TRBC2).
[0569] В некоторых вариантах осуществления эндогенный TCR Cα кодируется геном TRAC (номенклатура IMGT). Иллюстративная последовательность локуса гена константного домена альфа-цепи Т-клеточного рецептора человека (TRAC) представлена в SEQ ID NO: 348 (Эталонная последовательность NCBI: NG_001332.3, TRAC). В некоторых вариантах осуществления генетическое нарушение подвергается направленному воздействию в, вблизи или в пределах локуса TRAC. В конкретных вариантах осуществления генетическое нарушение подвергается направленному воздействию в, вблизи или в пределах открытой рамки считывания локуса TRAC. В некоторых вариантах осуществления генетическое нарушение подвергается направленному воздействию в, вблизи или внутри открытой рамки считывания, которая кодирует константный домен TCRα. В некоторых вариантах осуществления генетическое разрушение подвергается направленному воздействию в, вблизи или внутри локуса, содержащего последовательность нуклеиновой кислоты, представленной в SEQ ID NO: 348, или последовательность, имеющую, по меньшей мере, 70%, 75%, 80%, 85% 90%, 95%, 97%, 98%, 99%, 99,5% или 99,9% идентичности последовательностей со всей или частью этой последовательности, например, по меньшей мере 500, 1000, 1500, 2000, 2500, 3000, 3500, или 4000 непрерывно расположенных нуклеотидов последовательности нуклеиновой кислоты, представленной в SEQ ID NO: 348.
[0570] У людей иллюстративный геномный локус TRAC содержит открытую рамку считывания, которая содержит 4 экзона и 3 интрона. Иллюстративный транскрипт мРНК TRAC может охватывать последовательность, соответствующую координатам хромосомы 14: 22,547,506-22,552,154, на прямой цепи на основании версии генома человека GRCh38 (UCSC Genome Browser on Human Dec. 2013 (GRCh38/hg38) Assembly). В Таблице 13 приведены координаты экзонов и интронов открытых рамок считывания и нетранслируемых областей транскрипта иллюстративного локуса TRAC человека.
Таблица 13 Координаты экзонов и интронов иллюстративного локуса TRAC человека (GRCh38, хромосома 14, прямая цепь).
[0571] В некоторых вариантах осуществления эндогенный TCR Cβ кодируется генами TRBC1 или TRBC2 (номенклатура IMGT). Иллюстративная последовательность локуса гена константного домена 1 (TRBC1) бета-цепи Т-клеточного рецептора человека представлена в SEQ ID NO: 349 (Эталонная последовательность NCBI: NG_001333.2, TRBC1); и иллюстративная последовательность локуса гена константного домена 2 бета-цепи Т-клеточного рецептора человека (TRBC2) представлена в SEQ ID NO: 1047 (Эталонная последовательность NCBI: NG_001333.2, TRBC2). В некоторых вариантах осуществления генетическое нарушение подвергается направленному воздействию в, вблизи или внутри локуса гена TRBC1. В конкретных вариантах осуществления генетическое нарушение подвергается направленному воздействию в, вблизи или внутри открытой рамки считывания локуса TRBC1. В некоторых вариантах осуществления генетическое нарушение подвергается направленному воздействию в, вблизи или внутри открытой рамки считывания, которая кодирует константный домен TCRβ. В некоторых вариантах осуществления генетическое нарушение подвергается направленному воздействию в, вблизи или внутри локуса, содержащего последовательность нуклеиновой кислоты, представленную в SEQ ID NO: 349, или последовательность, имеющую, по меньшей мере, 70%, 75%, 80%, 85% 90%, 95%, 97%, 98%, 99%, 99,5% или 99,9% идентичности последовательностей со всей или частью этой последовательности, например, по меньшей мере 500, 1000, 1500, 2000, 2500, 3000, 3500, или 4000 последовательно расположенных нуклеотидов последовательности нуклеиновой кислоты, представленной в SEQ ID NO: 349.
[0572] У человека иллюстративный геномный локус TRBC1 содержит открытую рамку считывания, которая содержит 4 экзона и 3 интрона. Иллюстративный транскрипт мРНК TRBC1 может охватывать последовательность, соответствующую координатам хромосомы 7: 142,791,694-142,793,368, на прямой цепи на основании версии генома человека GRCh38 (UCSC Genome Browser on Human Dec. 2013 (GRCh38/hg38) Assembly). В Таблице 14 приведены координаты экзонов и интронов открытых рамок считывания и нетранслируемых областей транскрипта иллюстративного локуса TRBC1 человека.
Таблица 14 Координаты экзонов и интронов иллюстративного локуса TRBC1 человека (GRCh38, хромосома 7, прямая цепь).
[0573] В конкретных вариантах осуществления генетическое нарушение подвергается направленному воздействию в, вблизи или внутри локуса TRBC2. В конкретных вариантах осуществления генетическое нарушение подвергается направленному воздействию в, вблизи или внутри открытой рамки считывания локуса TRBC2. В некоторых вариантах осуществления генетическое нарушение подвергается направленному воздействию в, вблизи или внутри открытой рамки считывания, которая кодирует константный домен TCRβ. В некоторых вариантах осуществления генетическое нарушение подвергается направленному воздействию в, вблизи или внутри локуса, содержащего последовательность нуклеиновой кислоты, представленную в SEQ ID NO: 1047, или последовательность, имеющую, по меньшей мере, 70%, 75%, 80%, 85% 90%, 95%, 97%, 98%, 99%, 99,5% или 99,9% идентичности последовательностей со всей или частью этой последовательности, например, по меньшей мере 500, 1000, 1500, 2000, 2500, 3000, 3500, или 4000 последовательно расположенных нуклеотидов последовательности нуклеиновой кислоты, представленной в SEQ ID NO: 1047.
[0574] У человека иллюстративный геномный локус TRBC2 содержит открытую рамку считывания, которая содержит 4 экзона и 3 интрона. Иллюстративный транскрипт мРНК TRBC2 может охватывать последовательность, соответствующую координатам хромосомы 7: 142,801,041-142,802,748, на прямой цепи на основании версии генома человека GRCh38 (UCSC Genome Browser on Human Dec. 2013 (GRCh38/hg38) Assembly). В Таблице 15 приведены координаты экзонов и интронов открытых рамок считывания и нетранслируемых областей транскрипта иллюстративного локуса TRBC2 человека.
Таблица 15 Координаты экзонов и интронов прмерного локуса TRBC2 человека (GRCh38, хромосома 7, прямая цепь).
[0575] В некоторых вариантах осуществления ген(гены), являющиеся мишенью для нарушения или нокаута, находятся в одном или вблизи одного или более из локусов TRAC, TRBC1 и/или TRBC2. В некоторых вариантах осуществления ген TRAC нокаутирован. В некоторых вариантах осуществления ген TRBC1 нокаутирован. В некоторых вариантах осуществления ген TRBC2 нокаутирован. В некоторых вариантах осуществления нокаутированы гены TRAC и TRBC1. В некоторых вариантах осуществления нокаутированы ген TRAC и ген TRBC2. В некоторых вариантах осуществления ген TRAC и оба гена TRBC1 и TRBC2 нокаутируют, например, направленно воздействуя на последовательность, которая является консервативной между TRBC1 и TRBC2.
[0576] В некоторых вариантах осуществления уменьшение или предотвращение экспрессии эндогенного TCR может привести к снижению риска или вероятности неправильного связывания между цепями сконструированного TCR и эндогенного TCR, создавая тем самым новый TCR, который потенциально может привести к более высокому риску нежелательного или непреднамеренного распознавания антигена и/или к побочным эффектам, и/или может снизиться уровень экспрессии целевого экзогенного TCR. В некоторых аспектах уменьшение или предотвращение эндогенной экспрессии TCR может увеличивать экспрессию сконструированного TCR в клетках по сравнению с клетками, в которых экспрессия TCR не снижается или не предотвращается, например, увеличивается в 1,5 раза, в 2 раза, в 3 раза, в 4 раза, в 5 раз или более. Например, в некоторых случаях субоптимальная экспрессия сконструированного или рекомбинантного TCR может происходить из-за конкуренции с эндогенным TCR и/или с TCR, содержащим неправильно спаренные цепи, за инвариантные сигнальные молекулы CD3, которые участвуют в обеспечении возможности экспрессии комплекса на клеточной поверхности.
[0577] В некоторых вариантах осуществления уменьшение, удаление, устранение, нокаут или нарушение включают использование одного или более агентов, способных вводить генетическое нарушение, расщепление, двухцепочечный разрыв (DSB) и/или одноцепочечный разрыв в сайте-мишени в геномной ДНК, что приводит к уменьшению, удалению, устранению, нокауту или нарушению после репарации различными механизмами репарации клеточной ДНК.
[0578] В некоторых вариантах осуществления один или более агентов, способных к введению расщепления, включают ДНК-связывающий белок или ДНК-связывающую нуклеиновую кислоту, которая специфически связывается или гибридизуется с сайтом-мишенью в геноме, например, в генах TRAC и/или TRBC. В некоторых аспектах направленное расщепление, например разрыв ДНК, эндогенных генов, кодирующих TCR, достигается с использованием белка или нуклеиновой кислоты, связанных или образующих комплекс с нуклеазой для редактирования гена, как например в химерном или гибридном белке. В некоторых вариантах осуществления один или более агентов, способных к введению расщепления, включают слитый белок, содержащий белок, нацеленный на ДНК, и нуклеазу или РНК-направляемую нуклеазу.
[0579] В некоторых вариантах осуществления восстановление, удаление, устранение, нокаут или нарушение осуществляются методами редактирования генов, такими как использование нуклеазы цинкового пальца (ZFN), TALEN или системы CRISPR/Cas вместе со сконструированной одиночной гид-РНК, которая расщепляет ген TCR. В некоторых вариантах осуществления снижение экспрессии эндогенного TCR осуществляют с использованием ингибирующей молекулы нуклеиновой кислоты против нуклеиновых кислот-мишеней, кодирующих специфические TCR (например, TCR-α и TCR-β). В некоторых вариантах осуществления ингибирующая нуклеиновая кислота представляет собой или содержит или кодирует короткую интерферирующую РНК (киРНК), микроРНК-адаптированную кшРНК, короткую шпилечную РНК (кшРНК), шпилечную киРНК, микроРНК (миРНК-предшественник) или микроРНК (миРНК). Иллюстративные способы снижения или предотвращения эндогенной экспрессии TCR известны в данной области, см., например, Патент США № 9273283; публикацию США № US2014/0301990; и публикацию PCT № WO2015/161276.
[0580] В некоторых вариантах осуществления агент, способный к введению направленного расщепления, включает различные компоненты, такие как слитый белок, содержащий ДНК-нацеленный белок и нуклеазу или РНК-направляемую нуклеазу. В некоторых вариантах осуществления направленное расщепление осуществляют с использованием молекулы, нацеленной на ДНК, которая включает ДНК-связывающий белок, такой как один или более белков цинкового пальца (ZFP), или эффекторы, подобные активатору транскрипции (TALE), слитые с нуклеазой, такой как как эндонуклеаза. В некоторых вариантах осуществления направленное расщепление осуществляют с использованием РНК-направляемых нуклеаз, таких как система нуклеазы Cas (включая Cas и/или Cfp1), ассоциированной с нуклеиновой кислотой CRISPR (короткие палиндромные повторы, регулярно расположенные группами). В некоторых вариантах осуществления направленное расщепление осуществляют с использованием агентов, способных вводить расщепление, таких как специфичные для последовательности или направленные нуклеазы, включая ДНК-связывающие направленные нуклеазы и нуклеазы редактирования генов, такие как нуклеазы цинкового пальца (ZFN) и эффекторные нуклеазы, подобные активатору транскрипции (TALEN) и РНК-направляемые нуклеазы, такие как система CRISPR-ассоциированной нуклеазы (Cas), специально сконструированная и/или предназначенная для нацеливания по меньшей мере на один сайт-мишень (сайты), последовательность гена или его часть.
1. Сконструированные нуклеазы
[0581] В некоторых вариантах осуществления один или более агентов специфически нацелены по меньшей мере на один сайт-мишень (сайты), например, на последовательности или вблизи генов TRAC и/или TRBC. В некоторых вариантах осуществления агент содержит комбинацию ZFN, TALEN или CRISPR/Cas9, которая специфически связывается, распознает или гибридизуется с сайтом-мишенью (сайтами). В некоторых вариантах осуществления система CRISPR/Cas9 включает сконструированную crРНК/tracr РНК («одиночная направляющая РНК») для направления специфического расщепления. В некоторых вариантах осуществления агент содержит нуклеазы на основе системы Argonaute (например, из T. thermophilus, известного как TtAgo) (Swarts et al. (2014) Nature 507 (7491): 258-261).
[0582] Белки цинкового пальца (ZFP), эффекторы, подобные активатору транскрипции (TALE), и домены, связывающие систему CRISPR, могут быть «сконструированы» для связывания с предварительно определенной нуклеотидной последовательностью, например, посредством конструирования (изменения одной или более аминокислот) спиральной области распознавания природного белка ZFP или TALE. Сконструированные ДНК-связывающие белки (ZFP или TALE) представляют собой белки, которые не встречаются в природе. Рациональные критерии для проектирования включают применение правил замещения и компьютеризированных алгоритмов для обработки информации в базе данных, хранящей информацию о существующих конструкциях ZFP и/или TALE и данных о связывании. Смотри, например, Патенты США No. 6140081; 6453242; и 6534261; см. также WO 98/53058; WO 98/53059; WO 98/53060; WO 02/016536 и WO 03/016496 и Публикацию США № 20110301073. Иллюстративные ZFN, TALE и TALEN описаны, например, в Lloyd et al., Frontiers in Immunology, 4 (221): 1-7 (2013).
[0583] В некоторых вариантах осуществления сконструированный белок цинкового пальца, белок TALE или система CRISPR/Cas не присутствуют в природе и продукция которых обусловлена главным образом эмпирическим процессом, таким как фаговый дисплей, ловушка взаимодействия или гибридный отбор. См., например, Патенты США № 5789538; Патенты США 5925523; Патенты США № 6007988; Патенты США № 6013453; Патенты США № 6200759; WO 95/19431; WO 96/06166; WO 98/53057; WO 98/54311; WO 00/27878; WO 01/60970; WO 01/88197 и WO 02/099084.
[0584] В некоторых вариантах осуществления гены TRAC и/или TRBC могут подвергаться направленному воздействию расщепления с помощью сконструированных ZFN. Иллюстративные ZFN, которые нацелены на эндогенные гены Т-клеточных рецепторов (TCR), включают те, которые описаны, например, в US 2015/0164954, US 2011/0158957, US 2015/0056705, US 8956828 и Torikawa et al. (2012) Blood 119: 5697-5705, раскрытия которых включены в качестве ссылки в полном объеме.
[0585] В некоторых вариантах осуществления гены TRAC и/или TRBC могут подвергаться направленному воздействию расщепления с помощью сконструированных TALEN. Иллюстративные TALEN, которые нацелены на эндогенные гены Т-клеточных рецепторов (TCR), включают гены, описанные, например, в WO 2017/070429, WO 2015/136001, US20170016025 и US20150203817, раскрытия которых включены в качестве ссылки в полном объеме.
2. CRISPR-связанные методы
[0586] В некоторых вариантах осуществления гены TRAC и/или TRBC могут подвергаться направленному воздействию расщепления с использованием коротких палиндромных повторов, регулярно расположенных группами (CRISPR) и CRISPR-ассоциированных (Cas) белков. См. Sander and Joung, (2014) Nature Biotechnology, 32 (4): 347-355. В некоторых вариантах осуществления «система CRISPR» в совокупности относится к транскриптам и другим элементам, участвующим в экспрессии или управлении активностью CRISPR-ассоциированных («Cas») генов, включая последовательности, кодирующие ген Cas, tracr (трансактивирующий CRISPR) последовательность (например, tracrРНК или активная частичная tracrРНК), последовательность tracr-mate (включающая «прямой повтор» и tracrРНК-процессированный частичный прямой повтор в контексте эндогенной системы CRISPR), направляющая последовательность (также называемая «спейсер» в контексте эндогенной системы CRISPR) и/или другие последовательности и транскрипты из локуса CRISPR.
[0587] В некоторых аспектах нуклеаза CRISPR/Cas или система нуклеазы CRISPR/Cas включает некодирующую гид-РНК (гРНК), которая специфически связывается с ДНК, и белок Cas (например, Cas9) с функциональностью нуклеазы.
[0588] В некоторых вариантах осуществления редактирование гена приводит к вставке или удалению в целевом локусе или «нокауту» целевого локуса и устранению экспрессии кодируемого белка. В некоторых вариантах осуществления редактирование гена достигается посредством негомологичного концевого присоединения (NHEJ) с использованием системы CRISPR/Cas9. В некоторых вариантах осуществления одну или более молекул гид-РНК (гРНК) можно использовать с одной или более нуклеазой Cas9, никазой Cas9, ферментативно неактивной Cas9 или их вариантами. Иллюстративные особенности молекулы (молекул) гРНК и молекулы (молекул) Cas9 описаны ниже.
[0589] В некоторых вариантах осуществления система нуклеазы CRISPR/Cas содержит, по меньшей мере, одно из: гид-РНК (гРНК), имеющую нацеливающий домен, который комплементарен сайту-мишени гена TRAC; гРНК, имеющую нацеливающий домен, который комплементарен сайту-мишени одного или обоих из генов TRBC1 и TRBC2; или, по меньшей мере, одну нуклеиновую кислоту, кодирующую гРНК.
[0590] В некоторых вариантах осуществления направляющая последовательность, например гид-РНК, представляет собой любые полинуклеотидные последовательности, содержащие по меньшей мере часть последовательности, например, нацеливающий домен, который обладает достаточной комплементарностью с последовательностью сайта-мишени, такой как сайт-мишень в TRAC, TRBC1 и/или генах TRBC2 у человека для гибридизации с последовательностью-мишенью в сайте-мишени и специфического связывания прямой последовательности комплекса CRISPR с последовательностью-мишенью. В некоторых вариантах осуществления в контексте образования комплекса CRISPR «сайт-мишень» (также известный как «положение-мишень», «ДНК-последовательность-мишень» или «локализация-мишень») может относиться к последовательности, для которой сконструирована направляющая последовательность, обладающая комплементарностью, где гибридизация между последовательностью-мишенью и доменом, например, нацеливающим доменом гид-РНК способствует образованию комплекса CRISPR. Полная комплементарность не обязательно требуется при условии, что существует достаточная комплементарность, чтобы вызвать гибридизацию и способствовать образованию комплекса CRISPR. В некоторых вариантах осуществления направляющая последовательность выбирается для уменьшения степени вторичной структуры в направляющей последовательности. Вторичная структура может быть определена с помощью любого подходящего алгоритма сворачивания полинуклеотидов.
[0591] В некоторых аспектах фермент CRISPR (например, нуклеаза Cas9) в комбинации с (и необязательно в комплексе с) направляющей последовательностью доставляется в клетку. Например, один или более элементов системы CRISPR являются производными от системы CRISPR типа I, типа II или типа III. Например, один или более элементов системы CRISPR происходят из конкретного организма, содержащего эндогенную систему CRISPR, такого как Streptococcus pyogenes, Staphylococcus aureus или Neisseria meningitides.
[0592] В некоторых вариантах осуществления гид-РНК (гРНК), специфичная для сайта-мишени (например, TRAC, TRBC1 и/или TRBC2 у человека), используется РНК-управляемыми нуклеазами, например, Cas, для введения ДНК-разрыва в сайте-мишени или положении-мишени. Способы конструирования гРНК и иллюстративных нацеливающих доменов могут включать методы, описанные, например, в международной публикации PCT №. WO2015/161276. Нацеливающие домены могут быть включены в гРНК, которая используется для нацеливания нуклеаз Cas9 на сайт-мишень или положение-мишень.
[0593] Способы выбора и проверки последовательностей-мишеней, а также анализы ошибочного нацеливания описаны, например, в Mali et al., 2013 Science 339 (6121): 823-826; Hsu et al. Nat Biotechnol, 31 (9): 827-32; Fu et al., 2014, Nat Biotechnol, doi: 10.1038/nbt.2808. PubMed PMID: 24463574; Heigwer et al., 2014 Nat Methods 11 (2): 122-3. doi: 10.1038/nmeth.2812. PubMed PMID: 24481216; Bae et al., 2014 Bioinformatics PubMed PMID: 24463181; Xiao A et al., 2014 Bioinformatics PubMed PMID: 24389662. Поногеномная база данных гРНК для редактирования генома CRISPR, которая содержит иллюстративные последовательности одиночных гид-РНК (sgРНК), нацеленные на конститутивные экзоны генов в геноме человека или геноме мыши (см., например, genescript.com/gRNA-database.html; см. также Sanjana et al. (2014) Nat. Methods, 11: 783-4). В некоторых аспектах последовательность гРНК представляет собой или включает последовательность с минимальным нецелевым связыванием с нецелевым сайтом или положением.
a. Молекулы гид-РНК (гРНК)
[0594] В некоторых вариантах осуществления агент содержит гРНК, которая нацелена на область локусов TRAC, TRBC1 и/или TRBC2. «Молекула гРНК» относится к нуклеиновой кислоте, которая способствует специфическому нацеливанию или возвращению комплекса молекула гРНК/молекула Cas9 к нуклеиновой кислоте-мишени, такой как локус в геномной ДНК клетки. Молекулы гРНК могут быть мономолекулярными (имеют одну молекулу РНК), иногда называемыми в настоящем описании «химерными» гРНК, или модульными (содержат более чем одну и, как правило, две, отдельные молекулы РНК).
[0595] Несколько иллюстративных структур гРНК с указанными на них доменами представлены на ФИГ. 14А-14G. Не желая быть связанными теорией, касающейся трехмерной формы или внутри- или межцепочечных взаимодействий активной формы гРНК, области высокой комплементарности иногда показаны в виде дуплексов на ФИГ. 14A-14G и других изображениях, представленных в настоящем описании.
[0596] В некоторых случаях гРНК представляет собой мономолекулярную или химерную гРНК, содержащую от 5' к 3': нацеливающий домен, который комплементарен нуклеиновой кислоте-мишени, такой как последовательность из генов TRAC, TRBC1 и/или TRBC2 (кодирующие последовательности, представленные в SEQ ID NO: 348, 349 и 1047, соответственно); первый домен комплементарности; связывающий домен; второй домен комплементарности (который комплементарен первому домену комплементарности); проксимальный домен; и, необязательно, хвостовой домен.
[0597] В других случаях гРНК представляет собой модульную гРНК, содержащую первую и вторую цепи. В этих случаях первая цепь предпочтительно включает от 5' к 3': нацеливающий домен (который комплементарен нуклеиновой кислоте-мишени, такой как последовательность из генов TRAC, TRBC1 и/или TRBC2, кодирующая последовательность, представленная в SEQ ID NO: 348, 349 и 1047, соответственно) и первый домен комплементарности. Вторая цепь обычно включает от 5' к 3': необязательно, 5'-домен удлинения; второй домен комплементарности; проксимальный домен; и, необязательно, хвостовой домен.
[0598] В некоторых случаях эта гРНК представляет собой мономолекулярную или химерную гРНК, содержащую от 5' к 3': нацеливающий домен, который нацелен на сайт-мишень или положение-мишень, например последовательность из локуса TRAC (иллюстративная нуклеотидная последовательность гена TRAC человека локус представлена в SEQ ID NO: 348; Эталонная последовательность NCBI: NG_001332.3, TRAC; иллюстративная геномная последовательность, описанная в Таблице 13 настоящего документа); первый домен комплементарности; связывающий домен; второй домен комплементарности (который комплементарен первому домену комплементарности); проксимальный домен; и, необязательно, хвостовой домен. В некоторых случаях эта гРНК представляет собой мономолекулярную или химерную гРНК, содержащую от 5' к 3': нацеливающий домен, который нацелен на сайт-мишень или положение-мишень, например последовательность из локуса TRBC1 или TRBC2 (иллюстративная нуклеотидная последовательность локус гена TRBC1 человека, представленная в SEQ ID NO: 349; Эталонная последовательность NCBI: NG_001333.2, TRBC1; иллюстративная геномная последовательность, описанная в Таблице 14 настоящего документа; иллюстративная нуклеотидная последовательность локуса гена TRBC2 человека, представленная в SEQ ID NO: 1047; NCBI Эталонная последовательность: NG_001333.2, TRBC2; иллюстративная геномная последовательность описана в Таблице 15 настоящего документа); первый домен комплементарности; связывающий домен; второй домен комплементарности (который комплементарен первому домену комплементарности); проксимальный домен; и, необязательно, хвостовой домен.
[0599] В других случаях гРНК представляет собой модульную гРНК, содержащую первую и вторую цепи. В этих случаях первая цепь предпочтительно включает от 5' к 3': нацеливающий домен (который нацелен на сайт-мишень или положение-мишень, например, в последовательности из локуса TRAC (иллюстративная нуклеотидная последовательность локуса гена TRAC человека, представленная в SEQ ID NO: 348; Эталонная последовательность NCBI: NG_001332.3, TRAC; иллюстративная геномная последовательность, описанная в Таблице 13 настоящего документа), или локуса TRBC1 или TRBC2 (иллюстративная нуклеотидная последовательность локуса гена TRBC1 человека, представленная в SEQ ID NO: 349; эталонная последовательность NCBI: NG_001333.2, TRBC11; иллюстративная геномная последовательность, описанная в Таблице 14 настоящего документа; иллюстративная нуклеотидная последовательность локуса гена TRBC2 человека, представленная в SEQ ID NO: 1047; Эталонная последовательность NCBI: NG_001333.2, TRBC2), и первая домен комплементарности. Вторая цепь обычно включает от 5' к 3': необязательно, 5'-домен удлинения; второй домен комплементарности; проксимальный домен; и, необязательно, хвостовой домен.
[0600] Эти домены кратко обсуждаются ниже:
(1) Нацеливающий Домен
[0601] На ФИГ. 14A-14G приведены примеры размещения нацеливающих доменов.
[0602] Нацеливающий домен содержит нуклеотидную последовательность, которая комплементарна, например, по меньшей мере, на 80, 85, 90, 95, 98 или 99%, например полностью комплементарна, последовательности-мишени нуклеиновой кислоты-мишени. Цепь нуклеиновой кислоты-мишени, содержащая последовательность-мишень, называется в настоящем описании «комплементарной цепью» нуклеиновой кислоты-мишени. Руководство по выбору нацеливающих доменов можно найти, например, в Fu Y et al., Nat Biotechnol 2014 (doi: 10.1038/nbt.2808) и Sternberg SH et al., Nature 2014 (doi: 10.1038/nature13011).
[0603] Нацеливающий домен является частью молекулы РНК и, следовательно, будет содержать основание урацил (U), в то время как любая ДНК, кодирующая молекулу гРНК, будет содержать основание тимин (Т). Не желая быть связанными теорией, в одном варианте осуществления полагают, что комплементарность нацеливающего домена с последовательностью-мишенью вносит вклад в специфичность взаимодействия комплекса молекулы гРНК/молекулы Cas9 с нуклеиновой кислотой-мишенью. Понятно, что в паре нацеливающего домена и последовательности-мишени основания урацила в нацеливающем домене будут спариваться с основаниями аденина в последовательности-мишени. В одном варианте осуществления домен-мишень сам по себе содержит в направлении от 5' к 3' необязательный вторичный домен и коровый домен. В одном варианте осуществления коровый домен полностью комплементарен последовательности-мишени. В одном варианте осуществления нацеливающий домен имеет длину от 5 до 50 нуклеотидов. Цепь нуклеиновой кислоты-мишени, которой нацеливающий домен является комплементарным, называется в настоящем описании комплементарной цепью. Некоторые или все нуклеотиды домена могут иметь модификацию, например сделать ее менее восприимчивой к деградации, улучшить биосовместимость и т. д. В качестве неограничивающего примера, остов домена-мишени может быть модифицирован с помощью фосфоротиоат или другой модификации (модификаций). В некоторых случаях нуклеотид нацеливающего домена может содержать 2'-модификацию, например, 2-ацетилирование, например, 2'-метилирование, или другие модификации.
[0604] В различных вариантах осуществления нацеливающий домен имеет длину 16-26 нуклеотидов (т. е. он имеет длину 16 нуклеотидов или длину 17 нуклеотидов, или длину 18, 19, 20, 21, 22, 23, 24, 25 или 26 нуклеотидов).
(2) Иллюстративные нацеливающие домены
[0605] В некоторых вариантах осуществления, когда нокаутным положением-мишенью T-клеток является кодирующая область TRAC, например, ранняя кодирующая область, и более чем одна гРНК используется для разрывов положений, например, двух одноцепочечных разрывов или двух двухцепочечных разрывов, или комбинации из одноцепочечных и двухцепочечных разрывов, например, для создания одной или более вставок/делеций в последовательности нуклеиновой кислоты-мишени, каждая гид-РНК независимо выбрана из одной из, представленных в Таблицах 25A-G или Таблице 29 международной публикации PCT No. WO2015161276.
[0606] В другом варианте осуществления, когда нокаутным положением-мишенью T-клеток является кодирующая область TRAC, например, ранняя кодирующая область, и более чем одна гРНК используется для разрывов положений, например, двух одноцепочечных разрывов или двух двухцепочечных разрывов, или комбинации из одноцепочечных и двухцепочечных разрывов, например, для создания одной или более вставок/делеций в последовательности нуклеиновой кислоты-мишени, каждая гид-РНК независимо выбрана из одной из, представленных в Таблицах 25A-G или Таблице 29 международной публикации PCT No. WO2015161276, так что разрыв создается с эффективностью более 10%.
[0607] В другом варианте осуществления, когда нокаутным положением-мишенью T-клеток является кодирующая область TRBC, например, ранняя кодирующая область, и более чем одна гРНК используется для разрывов положений, например, двух одноцепочечных разрывов или двух двухцепочечных разрывов, или комбинации из одноцепочечных и двухцепочечных разрывов, например, для создания одной или более вставок/делеций в последовательности нуклеиновой кислоты-мишени, каждая гид-РНК независимо выбрана из одной из, представленных в Таблицах 26A-G или Таблице 27 международной публикации PCT No. WO2015161276.
[0608] В другом варианте осуществления, когда нокаутным положением-мишенью T-клеток является кодирующая область TRBC, например, ранняя кодирующая область, и более чем одна гРНК используется для разрывов положений, например, двух одноцепочечных разрывов или двух двухцепочечных разрывов, или комбинации из одноцепочечных и двухцепочечных разрывов, например, для создания одной или более вставок/делеций в последовательности нуклеиновой кислоты-мишени, каждая гид-РНК независимо выбрана из одной из, представленных в Таблицах 26A-G или Таблице 27 международной публикации PCT No. WO2015161276, так что разрыв создается с эффективностью более 10%.
[0609] В некоторых вариантах осуществления иллюстративная последовательность нацеливающего домена гид-РНК включает любую из описанных в международной публикации PCT No. WO2015161276. В некоторых вариантах осуществления иллюстративные последовательности гид-РНК описаны ниже со ссылкой на Таблицы, представленные в международной публикации PCT No. WO2015161276, содержание которой включено в настоящее описание в качестве ссылки в полном объеме.
[0610] В Таблице 25А международной публликации РСТ No. WO2015161276 представлены нацеливающие домены для нокаутирования гена TRAC с использованием S. pyogenes Cas9, выбранной в соответствии с параметрами первого выбора. Нацеливающие домены связываются в пределах первых 500 п.н. кодирующей последовательности в 3'-области относительно исходного кодона и имеют хорошую ортогональность. Здесь предполагается, что нацеливающий домен гибридизуется с доменом-мишенью путем комплементарного спаривания оснований. Любой из нацеливающих доменов в таблице можно использовать с молекулой S. pyogenes Cas9, которая генерирует двухцепочечный разрыв (нуклеаза Cas9) или одноцепочечный разрыв (никаза Cas9). В одном варианте осуществления двойное нацеливание используется для создания двух одноцепочечных разрывов на противоположных цепях ДНК с использованием никазы S. pyogenes Cas9 с двумя нацеливающими доменами, которые комплементарны противоположным цепям ДНК, например, гРНК, содержащая любой нацеливающий домен минус-цепи, может быть спарена с любой гРНК, содержащей нацеливающий домен плюс-цепи, при условии, что две гРНК ориентированы на ДНК так, что PAM обращены наружу, а расстояние между 5'-концами гРНК составляет 0-50 п.н. В одном варианте осуществления две гРНК используются для нацеливания двух нуклеаз Cas9 или двух никаз Cas9, например, гРНК с нацеливающим доменом из группы A может быть спарена с гРНК с нацеливающим доменом из группы B, как показано в Таблице 25-1 Международной публикации РСТ No. WO2015161276.
[0611] В Таблице 25B Международной публикации РСТ No. WO2015161276 представлены нацеливающие домены для нокаутирования гена TRAC с использованием S. pyogenes Cas9, выбранной в соответствии с параметрами второго выбора. Нацеливающие домены связываются в пределах первых 500 п.н. кодирующей последовательности в 3'-области относительно исходного кодона, и хорошая ортогональность не требуется. Здесь предполагается, что нацеливающий домен гибридизуется с доменом-мишенью путем комплементарного спаривания оснований. Любой из нацеливающих доменов в таблице можно использовать с молекулой S. pyogenes Cas9, которая генерирует двухцепочечный разрыв (нуклеаза Cas9) или одноцепочечный разрыв (никаза Cas9). В одном варианте осуществления двойное нацеливание используется для создания двух одноцепочечных разрывов на противоположных цепях ДНК с использованием никазы S. pyogenes Cas9 с двумя нацеливающими доменами, которые комплементарны противоположным цепям ДНК, например, гРНК, содержащая любой нацеливающий домен минус-цепи, может быть спарена с любой гРНК, содержащей нацеливающий домен плюс-цепи, при условии, что две гРНК ориентированы на ДНК так, что PAM обращены наружу, а расстояние между 5'-концами гРНК составляет 0-50 п.н.
[0612] В Таблице 25C Международной публикации РСТ No. WO2015161276 представлены нацеливающие домены для нокаутирования гена TRAC с использованием S. aureus Cas9, выбранные в соответствии с параметрами первого выбора. Нацеливающие домены были отобраны в пределах первых 500 п.н. кодирующей последовательности, имели высокий уровень ортогональности и содержали NNGRRT PAM. Здесь предполагается, что нацеливающий домен гибридизуется с доменом-мишенью путем комплементарного спаривания оснований. Любой из нацеливающих доменов в таблице можно использовать с молекулой S. aureus Cas9, которая генерирует двухцепочечный разрыв (нуклеаза Cas9) или одноцепочечный разрыв (никаза Cas9). В одном варианте осуществления двойное нацеливание используется для создания двух одноцепочечных разрывов на противоположных цепях ДНК с использованием никазы S. aureus Cas9 с двумя нацеливающими доменами, которые комплементарны противоположным цепям ДНК, например, гРНК, содержащая любой нацеливающий домен минус-цепи, может быть спарена с любой гРНК, содержащей нацеливающий домен плюс-цепи, при условии, что две гРНК ориентированы на ДНК так, что PAM обращены наружу, а расстояние между 5'-концами гРНК составляет 0-50 п.н. В одном варианте осуществления две гРНК используются для нацеливания двух нуклеаз Cas9 или двух никаз Cas9, например, гРНК с нацеливающим доменом из группы A может быть спарена с гРНК с нацеливающим доменом из группы B, как показано в Таблице 25-2 Международной публикации РСТ No. WO201516127.
[0613] В Таблице 25D Международной публикации РСТ No. WO2015161276 представлены нацеливающие домены для нокаутирования гена TRAC с использованием S. aureus Cas9, выбранного в соответствии с параметрами второго выбора. Нацеливающие домены были выбраны в пределах первых 500 п.н. кодирующей последовательности, уровень ортогональности не требовался, и содержали NNGRRT PAM. Здесь предполагается, что нацеливающий домен гибридизуется с доменом-мишенью путем комплементарного спаривания оснований. Любой из нацеливающих доменов в таблице можно использовать с молекулой S. aureus Cas9, которая генерирует двухцепочечный разрыв (нуклеаза Cas9) или одноцепочечный разрыв (никаза Cas9). В одном варианте осуществления двойное нацеливание используется для создания двух одноцепочечных разрывов на противоположных цепях ДНК с использованием никазы S. aureus Cas9 с двумя нацеливающими доменами, которые комплементарны противоположным цепям ДНК, например, гРНК, содержащая любой нацеливающий домен минус-цепи, может быть спарена с любой гРНК, содержащей нацеливающий домен плюс-цепи, при условии, что две гРНК ориентированы на ДНК так, что PAM обращены наружу, а расстояние между 5'-концами гРНК составляет 0-50 п.н.
[0614] В Таблице 25E Международной публикации РСТ No. WO2015161276 представлены нацеливающие домены для нокаутирования гена TRAC с использованием S. aureus Cas9, выбранного в соответствии с параметрами третьего выбора. Нацеливающие домены были выбраны в оставшейся части кодирующей последовательности в 3'-области и содержали NNGRRT PAM. Здесь предполагается, что нацеливающий домен гибридизуется с доменом-мишенью путем комплементарного спаривания оснований. Любой из нацеливающих доменов в таблице можно использовать с молекулой S. aureus Cas9, которая генерирует двухцепочечный разрыв (нуклеаза Cas9) или одноцепочечный разрыв (никаза Cas9). В одном варианте осуществления двойное нацеливание используется для создания двух одноцепочечных разрывов на противоположных цепях ДНК с использованием никазы S. aureus Cas9 с двумя нацеливающими доменами, которые комплементарны противоположным цепям ДНК, например, гРНК, содержащая любой нацеливающий домен минус-цепи, может быть спарена с любой гРНК, содержащей нацеливающий домен плюс-цепи, при условии, что две гРНК ориентированы на ДНК так, что PAM обращены наружу, а расстояние между 5'-концами гРНК составляет 0-50 п.н.
[0615] В Таблице 25F Международной публикации РСТ No. WO2015161276 представлены нацеливающие домены для нокаутирования гена TRAC с использованием N.meningitides Cas9, выбранные в соответствии с параметрами первого выбора. Нацеливающие домены связываются в пределах первых 500 п.н. кодирующей последовательности в 3'-области относительно исходного кодона и имеют хорошую ортогональность. Здесь предполагается, что нацеливающий домен гибридизуется с доменом-мишенью путем комплементарного спаривания оснований. Любой из нацеливающих доменов в таблице можно использовать с молекулой N. meningitidis Cas9, которая генерирует двухцепочечный разрыв (нуклеаза Cas9) или одноцепочечный разрыв (никаза Cas9). В одном варианте осуществления двойное нацеливание используется для создания двух одноцепочечных разрывов на противоположных цепях ДНК с использованием никазы N. meningitidis с двумя нацеливающими доменами, которые комплементарны противоположным цепям ДНК, например, гРНК, содержащая любой нацеливающий домен минус-цепи, может быть спарена с любой гРНК, содержащей нацеливающий домен плюс-цепи, при условии, что две гРНК ориентированы на ДНК так, что PAM обращены наружу, а расстояние между 5'-концами гРНК составляет 0-50 п.н.
[0616] В Таблице 25G Международной публикации РСТ No. WO2015161276 представлены нацеливающие домены для нокаутирования гена TRAC с использованием N. meningitidis Cas9, выбранные в соответствии с параметрами второго выбора. Нацеливающие домены связываются в пределах первых 500 п.н. кодирующей последовательности в 3'-области относительно исходного кодона, и хорошая ортогональность не требуется. Здесь предполагается, что нацеливающий домен гибридизуется с доменом-мишенью путем комплементарного спаривания оснований. Любой из нацеливающих доменов в таблице можно использовать с молекулой N. meningitidis Cas9, которая генерирует двухцепочечный разрыв (нуклеаза Cas9) или одноцепочечный разрыв (никаза Cas9). В одном варианте осуществления двойное нацеливание используется для создания двух одноцепочечных разрывов на противоположных цепях ДНК с использованием никазы N. meningitidis Cas9 с двумя нацеливающими доменами, которые комплементарны противоположным цепям ДНК, например, гРНК, содержащая любой нацеливающий домен минус-цепи, может быть спарена с любой гРНК, содержащей нацеливающий домен плюс-цепи, при условии, что две гРНК ориентированы на ДНК так, что PAM обращены наружу, а расстояние между 5'-концами гРНК составляет 0-50 п.н.
[0617] В Таблице 26А Международной публикации РСТ No. WO2015161276 представлены нацеливающие домены для нокаутирования гена TRBC с использованием S. pyogenes Cas9, выбранные в соответствии с параметрами первого выбора. Нацеливающие домены связываются в пределах первых 500 п.н. кодирующей последовательности в 3'-области относительно исходного кодона и имеют хорошую ортогональность. Здесь предполагается, что нацеливающий домен гибридизуется с доменом-мишенью путем комплементарного спаривания оснований. Любой из нацеливающих доменов в таблице можно использовать с молекулой S. pyogenes Cas9, которая генерирует двухцепочечный разрыв (нуклеаза Cas9) или одноцепочечный разрыв (никаза Cas9). В одном варианте осуществления двойное нацеливание используется для создания двух одноцепочечных разрывов на противоположных цепях ДНК с использованием никазы S. pyogenes Cas9 с двумя нацеливающими доменами, которые комплементарны противоположным цепям ДНК, например, гРНК, содержащая любой нацеливающий домен минус-цепи, может быть спарена с любой гРНК, содержащей нацеливающий домен плюс-цепи, при условии, что две гРНК ориентированы на ДНК так, что PAM обращены наружу, а расстояние между 5'-концами гРНК составляет 0-50 п.н. В одном варианте осуществления две гРНК используются для нацеливания двух нуклеаз Cas9 или двух никаз Cas9, например, гРНК с нацеливающим доменом из группы A может быть спарена с гРНК с нацеливающим доменом из группы B, как показано в Таблице 26-1 Международной публикации РСТ No. WO201516127.
[0618] В Таблице 26B Международной публикации РСТ No. WO2015161276 представлены нацеливающие домены для нокаутирования гена TRBC с использованием S. pyogenes Cas9, выбранные в соответствии с параметрами второго выбора. Нацеливающие домены связываются в пределах первых 500 п.н. кодирующей последовательности в 3'-области относительно исходного кодона, и хорошая ортогональность не требуется. Здесь предполагается, что нацеливающий домен гибридизуется с доменом-мишенью путем комплементарного спаривания оснований. Любой из нацеливающих доменов в таблице можно использовать с молекулой S. pyogenes Cas9, которая генерирует двухцепочечный разрыв (нуклеаза Cas9) или одноцепочечный разрыв (никаза Cas9). В одном варианте осуществления двойное нацеливание используется для создания двух одноцепочечных разрывов на противоположных цепях ДНК с использованием никазы S. pyogenes Cas9 с двумя нацеливающими доменами, которые комплементарны противоположным цепям ДНК, например, гРНК, содержащая любой нацеливающий домен минус-цепи, может быть спарена с любой гРНК, содержащей нацеливающий домен плюс-цепи, при условии, что две гРНК ориентированы на ДНК так, что PAM обращены наружу, а расстояние между 5'-концами гРНК составляет 0-50 п.н.
[0619] В Таблице 26C Международной публикации PCT No. В WO2015161276 представлены нацеливающие домены для нокаутирования гена TRBC с использованием S. aureus Cas9, выбранные в соответствии с параметрами первого выбора. Нацеливающие домены были отобраны в пределах первых 500 п.н. кодирующей последовательности, имели высокий уровень ортогональности и содержали N GRRT PAM. Здесь предполагается, что нацеливающий домен гибридизуется с доменом-мишенью путем комплементарного спаривания оснований. Любой из нацеливающих доменов в таблице можно использовать с молекулой S. aureus Cas9, которая генерирует двухцепочечный разрыв (нуклеаза Cas9) или одноцепочечный разрыв (никаза Cas9). В одном варианте осуществления двойное нацеливание используется для создания двух одноцепочечных разрывов на противоположных цепях ДНК с использованием никазы S. aureus Cas9 с двумя нацеливающими доменами, которые комплементарны противоположным цепям ДНК, например, гРНК, содержащая любой нацеливающий домен минус-цепи, может быть спарена с любой гРНК, содержащей нацеливающий домен плюс-цепи, при условии, что две гРНК ориентированы на ДНК так, что PAM обращены наружу, а расстояние между 5'-концами гРНК составляет 0-50 п.н. В одном варианте осуществления две гРНК используются для нацеливания двух нуклеаз Cas9 или двух никаз Cas9, например, гРНК с нацеливающим доменом из группы A может быть спарена с гРНК с нацеливающим доменом из группы B, как показано в Таблице 26-2 Международной публикации РСТ No. WO201516127.
[0620] В Таблице 26D Международной публикации РСТ No. WO2015161276 представлены нацеливающие домены для нокаутирования гена TRBC с использованием S. aureus Cas9, выбранные в соответствии с параметрами второго выбора. Нацеливающие домены были выбраны в пределах первых 500 п.н. кодирующей последовательности, уровень ортогональности не требовался, и содержали NNGRRT PAM. Здесь предполагается, что нацеливающий домен гибридизуется с доменом-мишенью путем комплементарного спаривания оснований. Любой из нацеливающих доменов в таблице можно использовать с молекулой S. aureus Cas9, которая генерирует двухцепочечный разрыв (нуклеаза Cas9) или одноцепочечный разрыв (никаза Cas9). В одном варианте осуществления двойное нацеливание используется для создания двух одноцепочечных разрывов на противоположных цепях ДНК с использованием никазы S. aureus Cas9 с двумя нацеливающими доменами, которые комплементарны противоположным цепям ДНК, например, гРНК, содержащая любой нацеливающий домен минус-цепи, может быть спарена с любой гРНК, содержащей нацеливающий домен плюс-цепи, при условии, что две гРНК ориентированы на ДНК так, что PAM обращены наружу, а расстояние между 5'-концами гРНК составляет 0-50 п.н.
[0621] В Таблице 26E Международной публикации РСТ No. WO2015161276 представлены нацеливающие домены для нокаутирования гена TRBC с использованием S. aureus Cas9, выбранные в соответствии с параметрами третьего выбора. Нацеливающие домены были выбраны в оставшейся части кодирующей последовательности в 3'-области и содержали NNGRRT PAM. Здесь предполагается, что нацеливающий домен гибридизуется с доменом-мишенью путем комплементарного спаривания оснований. Любой из нацеливающих доменов в таблице можно использовать с молекулой S. aureus Cas9, которая генерирует двухцепочечный разрыв (нуклеаза Cas9) или одноцепочечный разрыв (никаза Cas9). В одном варианте осуществления двойное нацеливание используется для создания двух одноцепочечных разрывов на противоположных цепях ДНК с использованием никазы S. aureus Cas9 с двумя нацеливающими доменами, которые комплементарны противоположным цепям ДНК, например, гРНК, содержащая любой нацеливающий домен минус-цепи, может быть спарена с любой гРНК, содержащей нацеливающий домен плюс-цепи, при условии, что две гРНК ориентированы на ДНК так, что PAM обращены наружу, а расстояние между 5'-концами гРНК составляет 0-50 п.н.
[0622] В Таблице 26F Международной публикации РСТ No. WO2015161276 представлены нацеливающие домены для нокаутирования гена TRBC с использованием N. meningitidis Cas9, выбранные в соответствии с параметрами первого выбора. Нацеливающие домены связываются в пределах первых 500 п.н. кодирующей последовательности в 3'-области относительно исходного кодона и имеют хорошую ортогональность. Здесь предполагается, что нацеливающий домен гибридизуется с доменом-мишенью путем комплементарного спаривания оснований. Любой из нацеливающих доменов в таблице можно использовать с молекулой N. meningitidis Cas9, которая генерирует двухцепочечный разрыв (нуклеаза Cas9) или одноцепочечный разрыв (никаза Cas9). В одном варианте осуществления двойное нацеливание используется для создания двух одноцепочечных разрывов на противоположных цепях ДНК с использованием никазы N. meningitidis с двумя нацеливающими доменами, которые комплементарны противоположным цепям ДНК, например, гРНК, содержащая любой нацеливающий домен минус-цепи, может быть спарена с любой гРНК, содержащей нацеливающий домен плюс-цепи, при условии, что две гРНК ориентированы на ДНК так, что PAM обращены наружу, а расстояние между 5'-концами гРНК составляет 0-50 п.н.
[0623] В Таблице 26G Международной публикации РСТ No. WO2015161276 представлены нацеливающие домены для нокаутирования гена TRBC с использованием N.meningitides Cas9, выбранные в соответствии с параметрами второго выбора. Нацеливающие домены связываются в пределах первых 500 п.н. кодирующей последовательности в 3'-области относительно исходного кодона, и хорошая ортогональность не требуется. Здесь предполагается, что нацеливающий домен гибридизуется с доменом-мишенью путем комплементарного спаривания оснований. Любой из нацеливающих доменов в таблице можно использовать с молекулой N. meningitidis Cas9, которая генерирует двухцепочечный разрыв (нуклеаза Cas9) или одноцепочечный разрыв (никаза Cas9). В одном варианте осуществления двойное нацеливание используется для создания двух одноцепочечных разрывов на противоположных цепях ДНК с использованием никазы N. meningitidis Cas9 с двумя нацеливающими доменами, которые комплементарны противоположным цепям ДНК, например, гРНК, содержащая любой нацеливающий домен минус-цепи, может быть спарена с любой гРНК, содержащей нацеливающий домен плюс-цепи, при условии, что две гРНК ориентированы на ДНК так, что PAM обращены наружу, а расстояние между 5'-концами гРНК составляет 0-50 п.н.
[0624] В некоторых вариантах осуществления последовательность-мишень (домен-мишень) находится в или вблизи локусов TRAC, TRBC1 и/или TRBC2, например, в любой части кодирующей последовательности TRAC, TRBC1 и/или TRBC2, представленной в SEQ ID NO: 348, 349 и 1047, соответственно. В некоторых вариантах осуществления нуклеиновая кислота-мишень, комплементарная нацеливающему домену, расположена в ранней кодирующей области представляющего интерес гена, такого как TRAC, TRBC1 и/или TRBC2. Нацеливание на раннюю кодирующую область может быть использовано для нокаутирования (то есть устранения экспрессии) представляющего интерес гена. В некоторых вариантах осуществления ранняя кодирующая область представляющего интерес гена включает в себя последовательность, следующую непосредственно за стартовым кодоном (например, ATG), или в пределах 500 п.н. стартового кодона (например, менее чем 500, 450, 400, 350, 300, 250). 200, 150, 100, 50 п.н., 40 п.н., 30 п.н., 20 п.н. или 10 п.н.). В конкретных примерах нуклеиновая кислота-мишень находится в пределах 200 п.н., 150 п.н., 100 п.н., 50 п.н., 40 п.н., 30 п.н., 20 п.н. или 10 п.н. относительно стартового кодона. В некоторых примерах нацеливающий домен гРНК комплементарен, например, по меньшей мере, на 80, 85, 90, 95, 98 или 99%, например, полностью комплементарен последовательности-мишени на нуклеиновой кислоте-мишени, такой как нуклеиновая кислота-мишень в локусах TRAC, TRBC1 и/или TRBC2.
[0625] В некоторых вариантах осуществления генетическое нарушение, например разрыв ДНК, подвергается направленному воздействию в или в непосредственной близости от начала кодирующей области (например, в ранней кодирующей оласти, например, в пределах 500 п.н. от стартового кодона или оставшейся кодирующей последовательности, например в 3'-области первых 500 п.н. от стартового кодона). В некоторых вариантах осуществления генетическое нарушение, например разрыв ДНК, подвергается направленному воздействию в ранней кодирующей области представляющего интерес гена, например TRAC, TRBC1 и/или TRBC2, включая последовательность, следующую непосредственно за сайтом старта транскрипции, в пределах первого экзона кодирующей последовательности или в пределах 500 п.н. от сайта старта транскрипции (например, менее чем 500, 450, 400, 350, 300, 250, 200, 150, 100 или 50 п.н.) или в пределах 500 п.н. от старт-кодона (например, менее чем 500, 450, 400, 350, 300, 250, 200, 150, 100 или 50 п.н.).
[0626] В некоторых вариантах осуществления сайт-мишень находится в экзоне эндогенного локуса TRAC, TRBC1 и/или TRBC2. В некоторых вариантах осуществления сайт-мишень находится в интроне эндогенного локуса TRAC, TRBC1 и/или TRBC2. В некоторых аспектах сайт-мишень находится в регуляторном или контрольном элементе, например, промоторе, 5 'нетранслируемой области (UTR) или 3' UTR, локусе TRAC, TRBC1 и/или TRBC2. В некоторых вариантах осуществления сайт-мишень находится в открытой рамке считывания эндогенного локуса TRAC, TRBC1 и/или TRBC2. В конкретных вариантах осуществления сайт-мишень находится в пределах экзона в открытой рамке считывания локуса TRAC, TRBC1 и/или TRBC2.
[0627] В конкретных вариантах осуществления генетическое нарушение, например разрыв ДНК, подвергается направленному воздействию в области или внутри открытой рамки считывания интересующего гена или локуса, например, TRAC, TRBC1 и/или TRBC2. В некоторых вариантах осуществления генетическое нарушение подвергается направленному воздействию в области или внутри интрона в открытой рамке считывания гена или локуса, представляющего интерес. В некоторых вариантах осуществления генетическое нарушение подвергается направленному воздействию внутри экзона в открытой рамке считывания гена или локуса, представляющего интерес.
[0628] В конкретных вариантах осуществления генетическое нарушение, например разрыв ДНК, подвергается направленному воздействию в области интрона или внутри него. В некоторых вариантах осуществления генетическое нарушение, например разрыв ДНК, подвергается направленному воздействию в области экзона или внутри него. В некоторых вариантах осуществления генетическое нарушение, например разрыв ДНК, подвергается направленному воздействию в области или внутри экзона представляющего интерес гена, например TRAC, TRBC1 и/или TRBC2.
[0629] В некоторых вариантах осуществления генетическое нарушение, например разрыв ДНК, подвергается направленному воздействию внутри экзона гена TRAC, открытой рамки считывания или локуса. В некоторых вариантах осуществления генетическое нарушение происходит внутри первого экзона, второго экзона, третьего экзона или четвертого экзона гена TRAC, открытой рамки считывания или локуса. В конкретных вариантах осуществления генетическое нарушение находится в первом экзоне гена TRAC, открытой рамки считывания или локуса. В некоторых вариантах осуществления генетическое нарушение находится внутри 500 пар оснований (п.н.) в 3'-области от 5'-конца первого экзона в гене TRAC, открытой рамке считывания или локусе. В конкретных вариантах осуществления генетическое нарушение находится между крайним 5'-нуклеотидом экзона 1 и в 5'-области от крайнего 3'-нуклеотида экзона 1. В некоторых вариантах осуществления генетическое нарушение находится в пределах 400 п.н., 350 п.н., 300 п.н., 250 п.н., 200 п.н., 150 п.н., 100 п.н. или 50 п.н. в 3'-области от 5'-конца первого экзона в гене TRAC, открытой рамки считывания или локуса. В конкретных вариантах осуществления генетическое нарушение находится от 1 п.н. до 400 п.н., от 50 до 300 п.н., от 100 до 200 п.н. или от 100 до 150 п.н. в 3'-области от 5'-конца первого экзона в гене TRAC, открытой рамке считывания, или локусе, каждое включительно. В некоторых вариантах осуществления генетическое нарушение находится в области между 100 п.н. и 150 п.н. в 3'-области от 5'-конца первого экзона в гене TRAC, открытой рамке считывания или локусе, включительно.
[0630] В конкретных вариантах осуществления генетическое нарушение, например разрыв ДНК, подвергается направленному воздействию внутри экзона гена TRBC, открытой рамки считывания или локуса, например TRBC1 и/или TRBC2. В некоторых вариантах осуществления генетическое нарушение находится внутри первого экзона, второго экзона, третьего экзона или четвертого экзона гена TRBC1 и/или TRBC2, открытой рамки считывания или локуса. В некоторых вариантах осуществления генетическое нарушение находится внутри первого экзоне гена TRBC1 и/или гена TRBC2, открытой рамки считывания или локуса. В некоторых вариантах осуществления генетическое нарушение находится внутри первого экзона, второго экзона, третьего экзона или четвертого экзона гена TRBC1 и/или TRBC2, открытой рамки считывания или локуса. В некоторых вариантах осуществления генетическое нарушение находится между крайним 5'-нуклеотидом экзона 1 и в 5'-области от крайнего 3'-нуклеотида экзона 1. В конкретных вариантах осуществления генетическое нарушение находится в первом экзоне гена TRBC, открытой рамки считывания или локуса. В некоторых вариантах осуществления генетическое нарушение находится в пределах 400 п.н., 350 п.н., 300 п.н., 250 п.н., 200 п.н., 150 п.н., 100 п.н. или 50 п.н. в 3'-области от 5'-конца первого экзона гена TRBC1 и/или TRBC2, открытой рамки считывания или локуса. В конкретных вариантах осуществления генетическое нарушение составляет от 1 до 400 п.н., от 50 до 300 п.н., от 100 до 200 п.н. или от 100 до 150 п.н. в 3'-области от 5'-конца первого экзона гена TRBC1 и/или TRBC2, открытой рамки считывания или локуса, каждый включительно. В некоторых вариантах осуществления генетическое нарушение находится между 100 п.н. и 150 п.н. в 3'-области от 5'-конца первого экзона в гене TRBC1 и/или TRBC2, в открытой рамке считывания или локусе, включительно.
[0631] В некоторых вариантах осуществления нацеливающий домен для нокаута или нокдауна TRAC, TRBC1 и/или TRBC2 представляет собой или содержит последовательность, выбранную из любой из SEQ ID NO: 1048, 1053, 1229-1315.
[0632] Иллюстративные нацеливающие домены, содержащиеся в гРНК для нацеливания на генетическое нарушение человеческого TRAC, TRBC1 или TRBC2, включают в себя те, которые описаны, например, в WO2015/161276, WO2017/193107, WO2017/093969, US2016/272999 и US2015/056705 или нацеливающий домен, который может связываться с нацеливающими последовательностями, описанными выше. Иллюстративные нацеливающие домены, содержащиеся в гРНК, для нацеливания на генетическое нарушение локуса TRAC человека с использованием нуклеазы Cas9, такой как S. pyogenes или S. aureus Cas9, могут включать любые из доменов, прдставленных в Таблице 16 ниже.
Таблица 16 Иллюстративные последовательности нацеливающего домена гРНК TRAC
[0633] Иллюстративные нацеливающие домены, содержащиеся в гРНК, для нацеливания на генетическое нарушение локуса TRBC1 или TRBC2 человека с использованием нуклеазы Cas9, такой как S. pyogenes или S. aureus Cas9, могут включать любые из доменов, представленных в Таблице 17 ниже.
Таблица 17 Иллюстративные последовательности нацеливающих доменов гРНК TRBC1 или TRBC2
[0634] В некоторых вариантах осуществления гРНК для нацеливания на TRAC, TRBC1 и/или TRBC2 может быть любой, которая описана в данном документе, или описана в другом месте, например, в WO2015/161276, WO2017/193107, WO2017/093969, US2016/272999 и US2015/056705, или нацеливающий домен, который может связываться с нацеливающими последовательностями, описанными выше. В некоторых вариантах осуществления последовательность, на которую нацелена гРНК CRISPR/Cas9 в локусе гена TRAC, представляет собой GAGAATCAAAATCGGTGAAT (SEQ ID NO: 1348) или ATTCACCGATTTTGATTCTC (SEQ ID NO: 1182). В некоторых вариантах осуществления последовательность, на которую нацелена гРНК CRISPR/Cas9 в локусах гена TRBC1 и/или TRBC2, представляет собой GGCCTCGGCGCTGACGATCT (SEQ ID NO: 1349) или AGATCGTCAGCGCCGAGGCC (SEQ ID NO: 1054). В некоторых вариантах осуществления последовательность нацеливающего домена гРНК для нацеливания на сайт-мишень в локусе гена TRAC представляет собой GAGAAUCAAAAUCGGUGAAU (SEQ ID NO: 1048). В некоторых вариантах осуществления последовательность нацеливающего домена гРНК для нацеливания на сайт-мишень в локусах гена TRBC1 и/или TRBC2 представляет собой GGCCUCGGCGCUGACGAUCU (SEQ ID NO: 1053).
[0635] В некоторых вариантах осуществления гРНК для нацеливания на локус гена TRAC может быть получена in vitro транскрипцией последовательности AGCGCTCTCGTACAGAGTTGGCATTATAATACGACTCACTATAGGGGAGAATCAAAATCGGTGAATGTTTTAGAGCTAGAAATAGCAAGTTAAAATAAGGCTAGTCCGTTATCAACTTGAAAAAGTGGCACCGAGTCGGTGCTTTTTTT (представленной в SEQ ID NO: 1350; выделена жирным шрифтом и подчеркнута часть, комплементарная сайту-мишени в локусе TRAC), или химически синтезирована, где гРНК имеет последовательность 5'- GAG AAU CAA AAU CGG UGA AUG UUU UAG AGC UAG AAA UAA UAG CAA GUU AAA AUA AGG CUA GUC CGU UAU CAA CUU GAA AAA GUG GCA CCG AGU CGG UGC UUU U-3 '(представленную в SEQ ID NO: 1351, см. Osborn et al., Mol Ther. 24 (3): 570-581 (2016). Другие иллюстративные последовательности гРНК или нацеливающие домены, содержащиеся в гРНК, и/или другие способы редактирования генов и/или нокаут, нацеленные на эндогенные гены TCR, например гены TRAC и/или TRBC, включают любые, описанные, например, в публикациях США № US2011/0158957, US2014/0301990, US2015/0098954, US2016/0208243; US2016/272999 и US2015/056705; Международной публикации PCT №. WO2014/191128, WO2015/136001, WO2015/161276, WO2016/069283, WO2016/016341, WO2017/193107 и WO2017/093969; и в Osborn et al. (2016) Mol. Ther. 24 (3): 570-581. Любой из известных способов может быть использован для генерации расщепления эндогенных генов, кодирующих домены или области TCR, и может быть использован в вариантах осуществления, предложенных в настоящем описании, например, для конструирования в клеточных линиях и/или в первичных Т-клетках.
[0636] В некоторых вариантах осуществления нацеливающие домены включают домены для нокаутирования генов TRAC, TRBC1 и/или TRBC2 с использованием S. pyogenes Cas9, S. aureus Cas9 или с использованием N. meningitidis Cas9.
[0637] В некоторых вариантах осуществления нацеливающие домены включают домены для нокаутирования генов TRAC, TRBC1 и/или TRBC2 с использованием S. pyogenes Cas9. Любой из нацеливающих доменов можно использовать с молекулой S. pyogenes Cas9, которая генерирует двухцепочечный разрыв (нуклеаза Cas9) или одноцепочечный разрыв (никаза Cas9).
[0638] В одном варианте осуществления двойное нацеливание используется для создания двух одноцепочечных разрывов на противоположных цепях ДНК с использованием никазы S. pyogenes Cas9 с двумя нацеливающими доменами, которые комплементарны противоположным цепям ДНК, например, гРНК, содержащая любой нацеливающий домен минус-цепи, может быть спарена с любой гРНК, содержащей нацеливающий домен плюс-цепи. В некоторых вариантах осуществления две гРНК ориентированы на ДНК так, что PAM обращены наружу, а расстояние между 5'-концами гРНК составляет 0-50 п.н. В одном варианте осуществления две гРНК используются для нацеливания двух нуклеаз Cas9 или двух никаз Cas9, например, с использованием пары комплекса молекулы Cas9/молекулы гРНК, направляемой двумя различными молекулами гРНК, для расщепления домена-мишени с двумя одноцепочечными разрывами на противоположных цепях домена-мишени. В некоторых вариантах осуществления две никазы Cas9 могут включать молекулу, обладающую активностью HNH, например молекулу Cas9, обладающую инактивированной активностью RuvC, например молекулу Cas9, имеющую мутацию в D10, например мутацию D10A, молекулу, обладающую активностью RuvC, например, молекулу Cas9, обладающую активностью HNH, например молекулу Cas9, имеющую мутацию в H840, например H840A, или молекулу, обладающую активностью RuvC, например молекулу Cas9, обладающую инактивированной активностью HNH, например молекулу Cas9, имеющую мутацию в N863, например, N863A. В некоторых вариантах осуществления каждая из двух гРНК находится в комплексе с никазой D10A Cas9.
(3) Первый домен комплементарности
[0639] На ФИГ. 14A-14G. приведены примеры первых доменов комплементарности. Первый домен комплементарности является комплементарным второму домену комплементарности, описанному ниже, и, как правило, обладает достаточной комплементарностью ко второму домену комплементарности для формирования дуплексной области по меньшей мере при некоторых физиологических условиях. Первый домен комплементарности обычно имеет длину от 5 до 30 нуклеотидов, и может иметь длину от 5 до 25 нуклеотидов, от 7 до 25 нуклеотидов, от 7 до 22 нуклеотидов, от 7 до 18 нуклеотидов или от 7 до 15 нуклеотидов. В различных вариантах осуществления первый домен комплементарности составляет 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24 или 25 нуклеотидов в длину.
[0640] Как правило, первый домен комплементарности не имеет точной комплементарности со вторым целевым доменом комплементарности. В некоторых вариантах осуществления первый домен комплементарности может иметь 1, 2, 3, 4 или 5 нуклеотидов, которые не комплементарны соответствующему нуклеотиду второго домена комплементарности. Например, сегмент из 1, 2, 3, 4, 5 или 6 (например, 3) нуклеотидов первого домена комплементарности может не спариваться в дуплексе и может образовывать недуплексную или беспетлевую область. В некоторых случаях неспаренная или беспетлевая область, например, беспетлевая область из 3 нуклеотидов, присутствует во втором домене комплементарности. Эта неспаренная область необязательно начинается с 1, 2, 3, 4, 5 или 6, например 4, нуклеотидов с 5'-конца второго домена комплементарности.
[0641] Первый домен комплементарности может включать в себя 3 субдомена, которые представляют собой в направлении от 5 'к 3': 5'-субдомен, центральный субдомен и 3'-субдомен. В одном варианте осуществления 5'-субдомен имеет длину 4-9, например 4, 5, 6, 7, 8 или 9 нуклеотидов. В одном варианте осуществления длина центрального субдомена составляет 1, 2 или 3, например, 1 нуклеотид. В одном варианте осуществления 3'-субдомен имеет длину от 3 до 25 нуклеотидов, например 4-22, 4-18 или 4-10, или 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24 или 25 нуклеотидов.
[0642] В некоторых вариантах осуществления первый и второй домены комплементарности при образовании дуплекса содержат 11 спаренных нуклеотидов, например, в последовательности гРНК (одна спаренная цепь подчеркнута, одна выделена жирным шрифтом): NNNNNNNNNNNNNNNNNNNNGUUUUAGAGCUAGAAAUAGCAAGUUAAAAUAAGGCUAGUCCGUUAUCAACUUGAAAAAGUGGCACCGAGUCGGUGC (SEQ ID NO:1316).
[0643] В некоторых вариантах осуществления первый и второй домены комплементарности при образовании дуплекса содержат 15 спаренных нуклеотидов, например, в последовательности гРНК (одна спаренная цепь подчеркнута, одна выделена жирным шрифтом): NNNNNNNNNNNNNNNNNNNNGUUUUAGAGCUAUGCUGAAAAGCAUAGCAAGUUAAAAUAAGGCUAGUCCGUUAUCAACUUGAAAAAGUGGCACCGAGUCGGUGC (SEQ ID NO:1317).
[0644] В некоторых вариантах осуществления первый и второй домены комплементарности при образовании дуплекса содержат 16 спаренных нуклеотидов, например, в последовательности гРНК (одна спаренная цепь подчеркнута, а одна выделена жирным шрифтом): NNNNNNNNNNNNNNNNNNNNGUUUUAGAGCUAUGCUGGAAACAGCAUAGCAAGUUAAAAUAAGGCUAGUCCGUUAUCAACUUGAAAAAGUGGCACCGAGUCGGUGC (SEQ ID NO:1318).
[0645] В некоторых вариантах осуществления первый и второй домены комплементарности при образовании дуплекса содержат 21 спаренных нуклеотидов, например, в последовательности гРНК (одна спаренная цепь подчеркнута, одна выделена жирным шрифтом): NNNNNNNNNNNNNNNNNNNNGUUUUAGAGCUAUGCUGUUUUGGAAACAAAACAGCAUAGCAAGUUAAAAUAAGGCUAGUCCGUUAUCAACUUGAAAAAGUGGCACCGAGUCGGUGC (SEQ ID NO: 1319).
[0646] В некоторых вариантах осуществления нуклеотиды заменяются для удаления поли-U-трактов, например, в последовательностях гРНК (замененные нуклеотиды подчеркнуты): NNNNNNNNNNNNNNNNNNNNGUAUUAGAGCUAGAAAUAGCAAGUUAAUAUAAGGCUAGUCCGUUAUCAACUUGAAAAAGUGGCACCGAGUCGGUGC (SEQ ID NO:1320); NNNNNNNNNNNNNNNNNNNNGUUUAAGAGCUAGAAAUAGCAAGUUUAAAUAAGGCUAGUCCGUUAUCAACUUGAAAAAGUGGCACCGAGUCGGUGC (SEQ ID NO:1321); и NNNNNNNNNNNNNNNNNNNNGUAUUAGAGCUAUGCUGUAUUGGAAACAAUACAGCAUAGCAAGUUAAUAUAAGGCUAGUCCGUUAUCAACUUGAAAAAGUGGCACCGAGUCGGUGC (SEQ ID NO:1322).
[0647] Первый домен комплементарности может иметь гомологию или быть получен из природного первого домена комплементарности. В одном варианте осуществления он имеет, по меньшей мере, 50% гомологии с первым доменом комплементарности, раскрытым в настоящем описании, например, с первым доменом комплементарности S. pyogenes, S. aureus, N. meningtidis или S. thermophilus.
[0648] Следует отметить, что один или более или даже все нуклеотиды первого домена комплементарности могут иметь модификацию в соответствии с обсуждаемым выше для нацеливающего домена.
(4) Связывающий домен
[0649] На ФИГ. 14A-14G приведены примеры связывающих доменов.
[0650] В мономолекулярной или химерной гРНК связывающий домен служит для связывания первого домена комплементарности со вторым доменом комплементарности мономолекулярной гРНК. Связывающий домен может связывать первый и второй домены комплементарности ковалентно или нековалентно. В одном варианте осуществления связь является ковалентной. В одном варианте осуществления связывающий домен ковалентно связывает первый и второй домены комплементарности, см., например, ФИГ. 14B-14E. В одном варианте осуществления связывающий домен представляет собой или содержит ковалентную связь, расположенную между первым доменом комплементарности и вторым доменом комплементарности. Обычно связывающий домен содержит один или более, например, 2, 3, 4, 5, 6, 7, 8, 9 или 10 нуклеотидов, но в различных вариантах осуществления линкер может содержать 20, 30, 40, 50 или даже 100 нуклеотидов в длину.
[0651] В модульных молекулах гРНК эти две молекулы ассоциированы в результате гибридизации доменов комплементарности, и связывающий домен может отсутствовать. Смотрите, например, ФИГ. 14А.
[0652] Широкий спектр связывающих доменов подходит для использования в мономолекулярных молекулах гРНК. Связывающие домены могут состоять из ковалентной связи или быть такими короткими, как один или более нуклеотидов, например, 1, 2, 3, 4 или 5 нуклеотидов в длину. В одном варианте осуществления связывающий домен имеет длину 2, 3, 4, 5, 6, 7, 8, 9, 10, 15, 20 или 25 или более нуклеотидов. В одном варианте осуществления длина связывающего домена составляет от 2 до 50, от 2 до 40, от 2 до 30, от 2 до 20, от 2 до 10 или от 2 до 5 нуклеотидов. В одном варианте осуществления связывающий домен гомологичен или происходит из встречающейся в природе последовательности, например, последовательности tracrРНК, которая находится в 5'-области относительно второго домена комплементарности. В одном варианте осуществления связывающий домен имеет по меньшей мере 50% гомологию со связывающим доменом, раскрытым в настоящем описании.
[0653] Как обсуждалось выше в связи с первым доменом комплементарности, некоторые или все нуклеотиды связывающего домена могут включать модификацию.
(5) 5'-Домен удлинения
[0654] В некоторых случаях модульная гРНК может содержать дополнительную последовательность в 5'-области относительно второго домена комплементарности, называемую здесь как 5'-домен удлинения, см., например, ABU. 14А. В одном варианте осуществления 5'-домен удлинения имеет длину 2-10, 2-9, 2-8, 2-7, 2-6, 2-5 или 2-4 нуклеотида. В одном варианте осуществления 5'-домен удлинения имеет длину 2, 3, 4, 5, 6, 7, 8, 9 или 10 или более нуклеотидов.
(6) Второй домен комплементарности
[0655] На ФИГ. 14А-14G приведены примеры вторых доменов комплементарности. Второй домен комплементарности является комплементарным первому домену комплементарности и, как правило, обладает достаточной комплементарностью ко второму домену комплементарности для формирования дуплексной области по меньшей мере при некоторых физиологических условиях. В некоторых случаях, например, как показано на ФИГ. 14A-14B, второй домен комплементарности может включать в себя последовательность, в которой отсутствует комплементарность с первым доменом комплементарности, например последовательность, которая не образует петли в дуплексной области.
[0656] Второй домен комплементарности может иметь длину от 5 до 27 нуклеотидов, а в некоторых случаях может быть длиннее, чем первая область комплементарности. Например, второй комплементарный домен может иметь длину от 7 до 27 нуклеотидов, длину от 7 до 25 нуклеотидов, длину от 7 до 20 нуклеотидов или длину от 7 до 17 нуклеотидов. В более общем случае, комплементарный домен может составлять 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25 или 26 нуклеотидов в длину.
[0657] В одном варианте осуществления второй домен комплементарности содержит 3 субдомена, которые в направлении от 5' к 3' представляют собой: 5'-субдомен, центральный субдомен и 3'-субдомен. В одном варианте осуществления 5'-субдомен составляет от 3 до 25, например, от 4 до 22, от 4 до 18, или от 4 до 10, или от 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24 или 25 нуклеотидов в длину. В одном варианте осуществления длина центрального субдомена составляет 1, 2, 3, 4 или 5, например 3 нуклеотида. В одном варианте осуществления 3'-субдомен имеет длину от 4 до 9, например, 4, 5, 6, 7, 8 или 9 нуклеотидов.
[0658] В одном варианте осуществления 5'-субдомен и 3'-субдомен первого домена комплементарности, соответственно, являются комплементарными, например полностью комплементарными, с 3'-субдоменом и 5'-субдоменом второго домена комплементарности.
[0659] Второй домен комплементарности может быть гомологичен или может быть получен из встречающегося в природе второго домена комплементарности. В одном варианте осуществления он имеет по меньшей мере 50% гомологию со вторым доменом комплементарности, раскрытым в настоящем описании, например, с первым доменом комплементарности S. pyogenes, S. aureus, N. meningtidis или S. thermophilus.
[0660] Некоторые или все нуклеотиды второго домена комплементарности могут иметь модификацию, например модификацию, раскрытую в разделе VIII настоящего описания.
(7) Проксимальный домен
[0661] На ФИГ. 14А-14G приведены примеры проксимальных доменов.
[0662] В одном варианте осуществления проксимальный домен имеет длину от 5 до 20 нуклеотидов. В одном варианте осуществления проксимальный домен может иметь гомологию с природным проксимальным доменом или может быть получен из него. В одном варианте осуществления он имеет, по меньшей мере, 50% гомологии с проксимальным доменом, раскрытым в настоящем описании, например, с проксимальным доменом S. pyogenes, S. aureus, N. meningtidis или S. thermophilus.
[0663] Некоторые или все нуклеотиды проксимального домена могут иметь модификацию в соответствии с описанным выше.
(8) Хвостовой домен
[0664] На ФИГ. 14А-14G приведены примеры хвостовых доменов.
[0665] Как видно из осмотра хвостовых доменов на ФИГ. 14А и ФИГ. 14B-14F, широкий спектр хвостовых доменов подходит для использования в молекулах гРНК. В различных вариантах осуществления хвостовой домен имеет длину 0 (отсутствует), 1, 2, 3, 4, 5, 6, 7, 8, 9 или 10 нуклеотидов. В некоторых вариантах осуществления нуклеотиды хвостового домена получены из или являются гомологичными последовательности из 5'-конца природного хвостового домена, см., например, ФИГ. 14D или 14E. Хвостовой домен также необязательно включает последовательности, которые комплементарны друг другу и которые, по меньшей мере, в некоторых физиологических условиях образуют дуплексную область.
[0666] Хвостовые домены могут иметь гомологию с природными проксимальными хвостовыми доменами или могут быть получены из них. В качестве неограничивающего примера, данный хвостовой домен в соответствии с различными вариантами осуществления настоящего изобретения может иметь по меньшей мере 50% гомологии с встречающимся в природе хвостовым доменом, раскрытым в настоящем описании, например, с хвостовым доменом S. pyogenes, S. aureus, N. meningtidis. или S. thermophilus.
[0667] В некоторых случаях хвостовой домен включает нуклеотиды на 3'-конце, которые связаны с методом in vitro или in vivo транскрипции. Когда промотор Т7 используется для in vitro транскрипции гРНК, этими нуклеотидами могут быть любые нуклеотиды, присутствующие до 3'-конца ДНК-матрицы. Когда промотор U6 используется для in vivo транскрипции, эти нуклеотиды могут представлять собой последовательность UUUUUU. Когда используются альтернативные промоторы pol-III, эти нуклеотиды могут представлять собой различное количество или основания урацила или могут включать альтернативные основания.
[0668] В качестве неограничивающего примера в различных вариантах осуществления проксимальный и хвостовой домены, взятые вместе, включают следующие последовательности:
AAGGCUAGUCCGUUAUCAACUUGAAAAAGUGGCACCGAGUCGGUGCU (SEQ ID NO: 1323), AAGGCUAGUCCGUUAUCAACUUGAAAAAGUGGCACCGAGUCGGUGGUGC (SEQ ID NO: 1324), AAGGCUAGUCCGUUAUCAACUUGAAAAAGUGGCACCGAGUCGGUGCGGAUC (SEQ ID NO: 1325), AAGGCUAGUCCGUUAUCAACUUGAAAAAGUG (SEQ ID NO: 1326), AAGGCUAGUCCGUUAUCA (SEQ ID NO: 1327), или AAGGCUAGUCCG (SEQ ID NO: 1328).
[0669] В одном варианте осуществления хвостовой домен содержит 3'-последовательность UUUUUU, например, если для транскрипции используется промотор U6.
[0670] В одном варианте осуществления хвостовой домен содержит 3'-последовательность UUUU, например, если для транскрипции используется промотор H1.
[0671] В одном варианте осуществления хвостовой домен содержит переменное количество 3'-U в зависимости, например, от сигнала терминации используемого промотора pol-III.
[0672] В одном варианте осуществления хвостовой домен содержит вариабельную 3'-последовательность, полученную из ДНК-матрицы, если используется промотор T7.
[0673] В одном варианте осуществления хвостовой домен содержит вариабельную 3'-последовательность, полученную из ДНК-матрицы, например, если используется в in vitro транскрипции для генерирования молекулы РНК.
[0674] В одном варианте осуществления хвостовой домен содержит вариабельную 3'-последовательность, полученную из ДНК-матрицы, например, если для управления транскрипцией используется промотор pol-II.
[0675] В одном варианте осуществления гРНК имеет следующую структуру:
5'[нацеливающий домен] - [первый домен комплементарности] - [связывающий домен] - [второй домен комплементарности] - [проксимальный домен] - [хвостовой домен] -3'
где нацеливающий домен содержит коровый домен и, необязательно, вторичный домен, и имеет длину от 10 до 50 нуклеотидов, первый домен комплементарности имеет длину от 5 до 25 нуклеотидов и, в одном варианте осуществления, имеет по меньшей мере 50, 60, 70, 80, 85, 90, 95, 98 или 99% гомологии с эталонным первым доменом комплементарности, раскрытым в настоящем описании, связывающий домен имеет длину от 1 до 5 нуклеотидов;
проксимальный домен имеет длину от 5 до 20 нуклеотидов и в одном варианте осуществления имеет гомологию по меньшей мере 50, 60, 70, 80, 85, 90, 95, 98 или 99% с раскрытым в настоящем описании эталонным проксимальным доменом; и
хвостовой домен отсутствует или нуклеотидная последовательность имеет длину от 1 до 50 нуклеотидов и в одном варианте осуществления имеет гомологию по меньшей мере 50, 60, 70, 80, 85, 90, 95, 98 или 99% с раскрытым в настоящем описании эталонным хвостовым доменом.
(9) Иллюстративные химерные гРНК
[0676] В одном из вариантов осуществления мономолекулярная или химерная гРНК содержит, предпочтительно от 5' к 3': нацеливающий домен, например, содержащий 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, или 26 нуклеотидов (которые комплементарны нуклеиновой кислоте-мишени); первый домен комплементарности; связывающий домен; второй домен комплементарности (который комплементарен первому домену комплементарности); проксимальный домен; и хвостовой домен, где (а) проксимальный и хвостовой домены, взятые вместе, содержат по меньшей мере 15, 18, 20, 25, 30, 31, 35, 40, 45, 49, 50 или 53 нуклеотида; (b) имеется по меньшей мере 15, 18, 20, 25, 30, 31, 35, 40, 45, 49, 50 или 53 нуклеотида в 3'-области относительно последнего нуклеотида второго домена комплементарности; или (c) имеется по меньшей мере 16, 19, 21, 26, 31, 32, 36, 41, 46, 50, 51 или 54 нуклеотида в 3'-области относительно последнего нуклеотида второго домена комплементарности, который комплементарен его соответствующему нуклеотиду первого домена комплементарности.
[0677] В одном варианте осуществления последовательность из (а), (b) или (с) имеет по меньшей мере 60, 75, 80, 85, 90, 95 или 99% гомологии с соответствующей последовательностью встречающейся в природе гРНК, или с гРНК, описанной в настоящем документе. В одном варианте осуществления проксимальный и хвостовой домены, взятые вместе, содержат по меньшей мере 15, 18, 20, 25, 30, 31, 35, 40, 45, 49, 50 или 53 нуклеотида. В одном варианте осуществления имеется по меньшей мере 15, 18, 20, 25, 30, 31, 35, 40, 45, 49, 50 или 53 нуклеотида в 3'-области относительно последнего нуклеотида второго домена комплементарности. [0684] В одном варианте осуществления имеется по меньшей мере 16, 19, 21, 26, 31, 32, 36, 41, 46, 50, 51 или 54 нуклеотида в 3'-области относительно последнего нуклеотида второго домена комплементарности, который комплементарен его соответствующему нуклеотиду первого домена комплементарности. В одном варианте осуществления нацеливающий домен содержит, имеет или состоит из 16, 17, 18, 19, 20, 21, 22, 23, 24, 25 или 26 нуклеотидов (например, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25 или 26 последовательных нуклеотидов), имеющих комплементарность с доменом-мишенью, например, нацеливающий домен составляет 16, 17, 18, 19, 20, 21, 22, 23, 24, 25 или 26 нуклеотидов в длину.
[0678] В одном варианте осуществления мономолекулярная или химерная молекула гРНК (содержащая нацеливающий домен, первый комплементарный домен, связывающий домен, второй комплементарный домен, проксимальный домен и, необязательно, хвостовой домен) содержит следующую последовательность, в которой нацеливающий домен обозначен как 20 N, но может быть любой последовательностью и длиной от 16 до 26 нуклеотидов, в которой за последовательностью гРНК следует 6 U, которые служат в качестве сигнала терминации для промотора U6, но которые могут отсутствовать или их может быть меньше: NNNNNNNNNNNNNNNNNNNNGUUUAGAGCUAGAAAUAGCAAGUUAAAUAAGGCUAGUCCGUUAUCAACUUGAAAAAGUGGCACCGAGUCGGUGCUUUUUUU (SEQ ID NO 1329). В одном варианте осуществления мономолекулярная или химерная молекула гРНК представляет собой молекулу гРНК S. pyogenes.
[0679] В некоторых вариантах осуществления мономолекулярная или химерная молекула гРНК (содержащая нацеливающий домен, первый комплементарный домен, связывающий домен, второй комплементарный домен, проксимальный домен и, необязательно, хвостовой домен) содержит следующую последовательность, в которой нацеливающий домен обозначен как 20 N, но может быть любой последовательностью и длиной от 16 до 26 нуклеотидов, в которой за последовательностью гРНК следуют 6 U, которые служат в качестве сигнала терминации для промотора U6, но которые могут отсутствовать или их может быть меньше: NNNNNNNNNNNNNNNNNNNNGUUUUAGUACUCUGGAAACAGAAUCUACUAAAACAAGGCAAAAUGCCGUGUUUUAUCUCGUCAACUUGUUGGCGCAGAGUUUUUUUU (SEQ ID NO: 1330). В одном варианте осуществления мономолекулярная или химерная молекула гРНК представляет собой молекулу гРНК S. aureus.
[0680] В некоторых вариантах осуществления нацеливающий домен в иллюстративной химерной гРНК представляет собой или содержит последовательность, выбранную из любой из SEQ ID NO: 1048, 1053, 1229-1315. В некоторых вариантах осуществления нацеливающий домен в иллюстративной химерной гРНК представляет собой или содержит последовательность, выбранную из любой из представленных в Таблице 16 или 17.
[0681] Последовательности и структуры иллюстративных химерных гРНК также показаны на ФИГ. 14А-14В.
(10) Иллюстративные Модульные гРНК
[0682] В одном варианте осуществления модульная гРНК содержит первую и вторую цепи. Первая цепь содержит, предпочтительно, от 5' к 3'; нацеливающий домен, например, содержащий 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25 или 26 нуклеотидов; первый домен комплементарности. Вторая цепь содержит, предпочтительно от 5' к 3': необязательно 5'-домен удлинения; второй домен комплементарности; проксимальный домен; и хвостовой домен, где: (а) проксимальный и хвостовой домен, взятые вместе, содержат по меньшей мере 15, 18, 20, 25, 30, 31, 35, 40, 45, 49, 50 или 53 нуклеотида; (b) имеется по меньшей мере 15, 18, 20, 25, 30, 31, 35, 40, 45, 49, 50 или 53 нуклеотида в 3'-области относительно последнего нуклеотида второго домена комплементарности; или (c) имеется по меньшей мере 16, 19, 21, 26, 31, 32, 36, 41, 46, 50, 51 или 54 нуклеотида в 3'-области относительно последнего нуклеотида второго домена комплементарности, который комплементарен его соответствующему нуклеотиду первого домена комплементарности.
[0683] В одном варианте осуществления последовательность из (а), (b) или (с) имеет по меньшей мере 60, 75, 80, 85, 90, 95 или 99% гомологии с соответствующей последовательностью встречающейся в природе гРНК, или с гРНК, описанной в настоящем документе. В одном варианте осуществления проксимальный и хвостовой домены, взятые вместе, содержат по меньшей мере 15, 18, 20, 25, 30, 31, 35, 40, 45, 49, 50 или 53 нуклеотида. В одном варианте осуществления имеется по меньшей мере 15, 18, 20, 25, 30, 31, 35, 40, 45, 49, 50 или 53 нуклеотида в 3'-области оносительно последнего нуклеотида второго домена комплементарности.
[0684] В одном варианте осуществления имеется по меньшей мере 16, 19, 21, 26, 31, 32, 36, 41, 46, 50, 51 или 54 нуклеотида в 3'-области относительно последнего нуклеотида второго домена комплементарности, который комплементарен его соответствующему нуклеотиду первого домена комплементарности.
[0685] В одном варианте осуществления нацеливающий домен имеет или состоит из 16, 17, 18, 19, 20, 21, 22, 23, 24, 25 или 26 нуклеотидов (например, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25 или 26 последовательных нуклеотидов), имеющих комплементарность с доменом-мишенью, например, нацеливающий домен имеет длину 16, 17, 18, 19, 20, 21, 22, 23, 24, 25 или 26 нуклеотидов.
[0686] В некоторых вариантах осуществления домен-мишень в иллюстративной модульной гРНК представляет собой или содержит последовательность, выбранную из любой из SEQ ID NO: 1048, 1053, 1229-1315. В некоторых вариантах осуществления нацеливающий домен в иллюстративной химерной гРНК представляет собой или содержит последовательность, выбранную из любой из представленных в Таблице 16 или 17.
b) Cas9
[0687] Молекулы Cas9 различных видов можно использовать в способах и композициях, описанных в настоящем документе. В то время как молекулы Cas9 S. pyogenes, S. aureus, N. meningitidis и S. thermophilus являются предметом большей части раскрытия настоящего описания, молекулы Cas9, полученные из или основанные на белках Cas9 других видов, перечисленных в настоящем описании, также могут быть использованы. Другими словами, хотя в большей части настоящего описания используются молекулы Cas9 S. pyogenes, S. aureus, N. meningitidis и S. thermophilus, молекулы Cas9 из других видов могут их заменить. Такие виды включают в себя: Acidovorax avenae, Actinobacillus pleuropneumoniae, Actinobacillus succinogenes, Actinobacillus suis, Actinomyces sp., Cycliphilusdenitrificans, Aminomonas paucivorans, Bacillus cereus, Bacillus smithii, Bacillus thuringiensis, Bacteroides sp., Blastopirellula marina, Bradyrhizobium sp., Brevibacillus laterosporus, Campylobacter coli, Campylobacter jejuni, Campylobacter lari, Candidatus puniceispirillum, Clostridium cellulolyticum, Clostridium perfringens, Corynebacterium accolens, Corynebacterium diphtheria, Corynebacterium matruchotii, Dinoroseobacter shibae, Eubacterium dolichum, Gammaproteobacterium, Gluconacetobacter diazotrophicus, Haemophilus parainfluenzae, Haemophilus sputorum, Helicobacter canadensis, Helicobacter cinaedi, Helicobacter mustelae, Ilyobacter polytropus, Kingella kingae, Lactobacillus crispatus, Listeria ivanovii, Listeria monocytogenes, Listeriaceae bacterium, Methylocystis sp., Methylosinus trichosporium, Mobiluncus mulieris, Neisseria bacilliformis, Neisseria cinerea, Neisseria flavescens, Neisseria lactamica, Neisseria meningitidis, Neisseria sp., Neisseria wadsworthii, Nitrosomonas sp., Parvibaculum lavamentivorans, Pasteurella multocida, Phascolarctobacterium succinatutens, Ralstonia syzygii, Rhodopseudomonas palustris, Rhodovulum sp., Simonsiella muelleri, Sphingomonas sp., Sporolactobacillus vineae, Staphylococcus aureus, Staphylococcus lugdunensis, Streptococcus sp., Subdoligranulum sp., Tistrella mobilis, Treponema sp., или Verminephrobacter eiseniae.
[0688] Молекула Cas9 или полипептид Cas9, как этот термин используется в настоящем описании, относится к молекуле или полипептиду, которые могут взаимодействовать с молекулой гРНК и, совместно с молекулой гРНК, располагается или локализуется в сайте, который содержит домен-мишень и PAM-последовательность. Молекула Cas9 и полипептид Cas9, как эти термины используются в настоящем описании, относятся к встречающимся в природе молекулам Cas9 и к сконструированным, измененным или модифицированным молекулам Cas9 или полипептидам Cas9, которые отличаются, например, по меньшей мере одним аминокислотным остатком, от эталонной последовательности, например, от наиболее сходной встречающейся в природе молекулы Cas9 или последовательности аминокислот, представленной в SEQ ID NO: 1331-1336, 1338, 1340 и 1341.
(1) Cas9 Домены
[0689] Кристаллические структуры были определены для двух различных встречающихся в природе бактериальных молекул Cas9 (Jinek et al., Science, 343 (6176): 1247997, 2014) и для Cas9 S. pyogenes с гид-РНК (например, синтетическое слияние crРНК и tracrРНК).) (Nishimasu et al., Cell, 156: 935-949, 2014; и Anders et al., Nature, 2014, doi: 10.1038/nature13579).
[0690] Встречающаяся в природе молекула Cas9 состоит из двух долей: доли распознавания (REC) и доли нуклеазы (NUC); каждая из которых дополнительно содержит домены, описанные в настоящем документе. Номенклатура доменов и нумерация аминокислотных остатков, охватываемых каждым доменом, используемым в этом раскрытии, являются такими, как описано в Nishimasu et al. Нумерация аминокислотных остатков относится к Cas9 из S. pyogenes.
[0691] Доля REC содержит богатую аргинином мостиковую спираль (BH), домен REC1 и домен REC2. Доля REC не имеет структурного сходства с другими известными белками, что указывает на то, что это специфический для Cas9 функциональный домен. Домен BH представляет собой длинную α-спиральную и богатую аргинином область, и содержит аминокислоты 60-93 последовательности Cas9 S. pyogenes. Домен REC1 важен для распознавания дуплекса повтор:антиповтр, например, гРНК или tracrРНК, и поэтому является критическим для активности Cas9 путем распознавания последовательности-мишени. Домен REC1 содержит два мотива REC1 в аминокислотах от 94 до 179 и от 308 до 717 последовательности Cas9 S. pyogenes. Эти два домена REC1, хотя и разделены доменом REC2 в линейной первичной структуре, собираются в третичную структуру, образуя домен REC1. Домен REC2 или его части также могут играть роль в распознавании дуплексов повтор:антиповтор. Домен REC2 содержит аминокислоты 180-307 последовательности Cas9 S. pyogenes.
[0692] Доля NUC содержит домен RuvC (также называемый в настоящем описании как RuvC-подобный домен), домен HNH (также называемый в настоящем описании как HNH-подобный домен) и PAM-взаимодействующий (PI) домен. Домен RuvC имеет структурное сходство с членами суперсемейства ретровирусных интеграз и расщепляет одну цепь, например некомплементарную цепь молекулы нуклеиновой кислоты-мишени. Домен RuvC собран из трех расщепленных мотивов RuvC (RuvC I, RuvCII и RuvCIII, которые часто обозначают в данной области доменом RuvCI или N-концевым доменом RuvC, доменом RuvCII и доменом RuvCIII) в аминокислотах 1-59, 718-769 и 909-1098, соответственно, последовательности Cas9 S. pyogenes. Подобно домену REC1, три мотива RuvC линейно разделены другими доменами в первичной структуре, однако в третичной структуре три мотива RuvC собираются и образуют домен RuvC. Домен HNH имеет структурное сходство с эндонуклеазами HNH и расщепляет одну цепь, например, комплементарную цепь молекулы нуклеиновой кислоты-мишени. Домен HNH находится между мотивами RuvC II-III и содержит аминокислоты 775-908 последовательности Cas9 S. pyogenes. Домен PI взаимодействует с PAM молекулы нуклеиновой кислоты-мишени и содержит аминокислоты 1099-1368 последовательности Cas9 S. pyogenes.
(a) RuvC-подобный домен и HNH-подобный домен
[0693] В одном варианте осуществления молекула Cas9 или полипептид Cas9 содержат HNH-подобный домен и RuvC-подобный домен. В одном варианте осуществления активность расщепления зависит от RuvC-подобного домена и HNH-подобного домена. Молекула Cas9 или полипептид Cas9, например, молекула eaCas9 или полипептид eaCas9, могут содержать один или более из следующих доменов: RuvC-подобный домен и HNH-подобный домен. В одном варианте осуществления молекула Cas9 или полипептид Cas9 представляют собой молекулу eaCas9 или полипептид eaCas9, а молекула eaCas9 или полипептид eaCas9 содержат RuvC-подобный домен, например, RuvC-подобный домен, описанный ниже, и/или HNH-подобный домен, например, HNH-подобный домен, описанный ниже.
(b) RuvC-подобные домены
[0694] В одном варианте осуществления RuvC-подобный домен расщепляет одну цепь, например некомплементарную цепь молекулы нуклеиновой кислоты-мишени. Молекула Cas9 или полипептид Cas9 могут включать более чем один RuvC-подобный домен (например, один, два, три или более RuvC-подобных домена). В одном варианте осуществления RuvC-подобный домен имеет длину по меньшей мере 5, 6, 7, 8 аминокислот, но не более чем 20, 19, 18, 17, 16 или 15 аминокислот. В одном варианте осуществления молекула Cas9 или полипептид Cas9 содержат N-концевой RuvC-подобный домен, составляющий примерно 10-20 аминокислот, например, примерно 15 аминокислот в длину.
(c) N-концевые RuvC-подобные домены
[0695] Некоторые встречающиеся в природе молекулы Cas9 содержат более чем один RuvC-подобный домен, причем расщепление зависит от N-концевого RuvC-подобного домена. Соответственно, молекулы Cas9 или полипептид Cas9 могут содержать N-концевой RuvC-подобный домен.
(d) Дополнительные RuvC-подобные домены
[0696] В дополнение к N-концевому RuvC-подобному домену молекула Cas9 или полипептид Cas9, например молекула eaCas9 или полипептид eaCas9, могут содержать один или более дополнительных RuvC-подобных доменов. В одном варианте осуществления молекула Cas9 или полипептид Cas9 могут содержать два дополнительных RuvC-подобных домена. Предпочтительно дополнительный RuvC-подобный домен имеет длину, составляющую по меньшей мере 5 аминокислот и, например, менее чем 15 аминокислот, например 5-10 аминокислот, например 8 аминокислот.
(e) HNH-подобные домены
[0697] В одном варианте осуществления HNH-подобный домен расщепляет одноцепочечный комплементарный домен, например, комплементарную цепь двухцепочечной молекулы нуклеиновой кислоты. В одном варианте осуществления HNH-подобный домен имеет длину, составляющую по меньшей мере 15, 20, 25 аминокислот, но не более чем 40, 35 или 30 аминокислот, например, от 20 до 35 аминокислот, например, от 25 до 30 аминокислот. Иллюстративные HNH-подобные домены описаны ниже.
[0698] В одном варианте осуществления HNH-подобный домен является способным к расщеплению.
[0699] В одном варианте осуществления HNH-подобный домен является неспособным к расщеплению.
(2) Активности Cas9
(a) Нуклеазная и хеликазная активности
[0700] В одном варианте осуществления молекула Cas9 или полипептид Cas9 способны расщеплять молекулу нуклеиновой кислоты-мишени. Обычно молекулы Cas9 дикого типа расщепляют обе цепи молекулы нуклеиновой кислоты-мишени. Молекулы Cas9 и полипептиды Cas9 могут быть сконструированы для изменения нуклеазного расщепления (или других свойств), например, для получения молекулы Cas9 или пептида Cas9, которые представляют собой никазу, или которые не обладают способностью расщеплять нуклеиновую кислоту-мишень. Молекула Cas9 или полипептид Cas9, которые способны расщеплять молекулу нуклеиновой кислоты-мищщени, обозначается в настоящем описании как молекула eaCas9 или полипептид eaCas9.
[0701] В одном варианте осуществления молекула eaCas9 или полипептид eaCas9 включают одну или более из следующих активностей:
активность никазы, то есть способность расщеплять одну цепь, например некомплементарную цепь или комплементарную цепь молекулы нуклеиновой кислоты;
двухцепочечную нуклеазную активность, то есть способность расщеплять обе цепи двухцепочечной нуклеиновой кислоты и создавать двухцепочечный разрыв, что в одном варианте осуществления представляет собой наличие двух никазных активностей;
эндонуклеазную активность;
экзонуклеазную активность; и
хеликазную активность, то есть способность раскручивать спиральную структуру двухцепочечной нуклеиновой кислоты.
[0702] В одном варианте осуществления ферментативно активная молекула или молекула eaCas9 или полипептид eaCas9 расщепляют обе цепи и приводят к двухцепочечному разрыву. В одном варианте осуществления молекула eaCas9 расщепляет только одну цепь, например, цепь, с которой гибридизуется гРНК, или цепь, комплементарную цепи, с которой гибридизуется гРНК. В одном варианте осуществления молекула eaCas9 или полипептид eaCas9 обладают активностью расщепления, ассоциированную с HNH-подобным доменом. В одном варианте осуществления молекула eaCas9 или полипептид eaCas9 обладают активностью расщепления, связанной с N-концевым RuvC-подобным доменом. В одном варианте осуществления молекула eaCas9 или полипептид eaCas9 обладают активностью расщепления, ассоциированную с HNH-подобным доменом, и активностью расщепления, ассоцциированную с N-концевым RuvC-подобным доменом. В одном варианте осуществления молекула eaCas9 или полипептид eaCas9 содержат активный или способный к расщеплению HNH-подобный домен и неактивный или неспособный к расщеплению N-концевой RuvC-подобный домен. В одном варианте осуществления молекула eaCas9 или полипептид eaCas9 содержат неактивный или неспособный к расщеплению HNH-подобный домен и активный или способный к расщеплению N-концевой RuvC-подобный домен.
[0703] Некоторые молекулы Cas9 или полипептиды Cas9 обладают способностью взаимодействовать с молекулой гРНК и совместно с молекулой гРНК локализуются в коровом домене-мишени, но неспособны расщеплять нуклеиновую кислоту-мишень или неспособны расщеплять в эффективной степени. Молекулы Cas9, не имеющие или не обладающие существенной активностью расщепления, обозначаются в настоящем описании как молекула eiCas9 или полипептид eiCas9. Например, молекула eiCas9 или полипептид eiCas9 могут не обладать активностью расщепления или иметь существенно меньшую, например, менее 20, 10, 5, 1 или 0,1% активности расщепления эталонной молекулы Cas9 или полипептида eiCas9, как измерено анализом, описанным в настоящем документе.
(b) Нацеливание и PAM
[0704] Молекула Cas9 или полипептид Cas9 представляет собой полипептид, который может взаимодействовать с молекулой гид-РНК (гРНК) и, совместно с молекулой гРНК, локализуется в сайте, который содержит домен-мишень и последовательность PAM.
[0705] В одном варианте осуществления способность молекулы eaCas9 или полипептида eaCas9 взаимодействовать и расщеплять нуклеиновую кислоту-мишень зависит от последовательности PAM. Последовательность PAM представляет собой последовательность в нуклеиновой кислоте-мишени. В варианте осуществления расщепление нуклеиновой кислоты-мишени происходит в 5'-области от последовательности PAM. Молекулы eaCas9 из разных видов бактерий могут распознавать разные последовательности мотивов (например, последовательности PAM). В одном варианте осуществления молекула eaCas9 S. pyogenes распознает последовательность мотива NGG, NAG, NGA и направляет расщепление последовательности нуклеиновой кислоты-мишени от 1 до 10, например от 3 до 5 пар оснований в 5'-области от этой последовательности. См., например, Mali et al., Science 2013; 339 (6121): 823-826. В одном варианте осуществления молекула eaCas9 S. thermophilus распознает последовательность мотива NGGNG и/или NNAGAAW (W=A или T) и направляет расщепление последовательности нуклеиновой кислоты-мишени от 1 до 10, например от 3 до 5 пар оснований в 5'-области от этих последовательностей. См., например, Horvath et al., Science 2010; 327 (5962): 167-170 и Deveau et al., J Bacteriol 2008; 190 (4): 1390-1400. В одном варианте осуществления молекула eaCas9 S. mutans распознает последовательность мотива NGG и/или NAAR (R=A или G)) и направляет расщепление коровой последовательности нуклеиновой кислоты-мишени от 1 до 10, например от 3 до 5 пар оснований в 5'-области от этой последовательности. См., например, Deveau et al., J Bacteriol 2008; 190 (4): 1390-1400. В одном варианте осуществления молекула eaCas9 S. aureus распознает последовательность мотива NNGRR (R=A или G) и направляет расщепление последовательности нуклеиновой кислоты-мишени от 1 до 10, например от 3 до 5 пар оснований в 5'-области от этой последовательности. В одном варианте осуществления молекула eaCas9 S. aureus распознает последовательность мотива NNGRRT (R=A или G) и направляет расщепление последовательности нуклеиновой кислоты-мишени от 1 до 10, например от 3 до 5 пар оснований в 5'-области от этой последовательности. В одном варианте осуществления молекула eaCas9 S. aureus распознает последовательность мотива NNGRRV (R=A или G) и направляет расщепление последовательности нуклеиновой кислоты-мишени от 1 до 10, например от 3 до 5 пар оснований в 5'-области от этой последовательности. В одном варианте осуществления молекула eaCas9 N. meningitidis распознает последовательность мотива NNNNGATT или NNNGCTT (R=A или G, V=A, G или C) и направляет расщепление последовательности нуклеиновой кислоты-мишени от 1 до 10, например от 3 до 5 пар оснований в 5'-области от этой последовательности. См., например, Hou et al., PNAS Early Edition 2013, 1-6. Способность молекулы Cas9 распознавать последовательность PAM может быть определена, например, с использованием анализа трансформации, описанного в Jinek et al., Science 2012 337: 816. В вышеупомянутых вариантах осуществления N может представлять собой любой нуклеотидный остаток, например, любой из A, G, C или T.
[0706] Как обсуждается в настоящем описании, молекулы Cas9 могут быть сконструированы для изменения специфичности PAM молекулы Cas9.
[0707] Иллюстративные природные молекулы Cas9 описаны в Chylinski et al., RNA Biology 2013 10: 5, 727-737. Такие молекулы Cas9 включают молекулы Cas9 бактериального семейства кластера 1-78.
[0708] Иллюстративные природные молекулы Cas9 включают молекулу Cas9 бактериального семейства кластера 1. Примеры включают молекулу Cas9: S. pyogenes (например, штамм SF370, MGAS10270, MGAS10750, MGAS2096, MGAS315, MGAS5005, MGAS6180, MGAS9429, NZ131 и SSI-1), S. thermophilus (например, штамм LMD-9), S pseudoporcinus (например, штамм SPIN 20026), S. mutans (например, штамм UA159, NN2025), S. macacae (например, штамм NCTC11558), S. gallolyticus (например, штамм UCN34, ATCC BAA-2069), S. equines (например, штамм ATCC 9812, MGCS 124), S. dysdalactiae (например, штамм GGS 124), S. bovis (например, штамм ATCC 700338), S. anginosus (например, штамм F0211), S. agalactiae (например, штамм NEM316, A909), Listeria monocytogenes (например, штамм F6854), Listeria innocua (L. innocua, например, штамм Clip11262), Enterococcus italicus (например, штамм DSM 15952) или Enterococcus faecium (например, штамм 1,231,408). Другой иллюстративной молекулой Cas9 является молекула Cas9 Neisseria meningitidis (Hou et al., PNAS Early Edition 2013, 1-6).
[0709] В одном варианте осуществления молекула Cas9 или полипептид Cas9, например молекула eaCas9 или полипептид eaCas9, содержат аминокислотную последовательность:
которая имеет 60%, 65%, 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98% или 99% гомологии; отличается не более чем на 2, 5, 10 15, 20, 30 или 40% аминокислотных остатков при сравнении;
отличается по меньшей мере на 1, 2, 5, 10 или 20 аминокислот, но не более чем на 100, 80, 70, 60, 50, 40 или 30 аминокислот; или идентична любой последовательности молекулы Cas9, описанной в настоящем документе, или последовательности молекулы Cas9 природного происхождения, например молекулы Cas9 из видов, перечисленных в настоящем описании или описанных в Chylinski et al., RNA Biology 2013 10: 5, 727-737; Hou et al., PNAS Early Edition 2013, 1-6; SEQ ID NO: 1331-1334 (S. mutans (SEQ ID NO:1331); S. pyogenes (SEQ ID NO:1332); S. thermophilus (SEQ ID NO:1333); L. innocua (SEQ ID NO:1334)). В одном варианте осуществления молекула Cas9 или полипептид Cas9 содержат одну или более из следующих активностей: активность никазы; активность двухцепочечного расщепления (например, эндонуклеазная и/или экзонуклеазная активность); хеликазная активность; или способность вместе с молекулой гРНК локализоваться с нуклеиновой кислотой-мишенью.
[0710] В одном варианте осуществления молекула Cas9 или полипептид Cas9 содержат аминокислотную последовательность консенсусной последовательности ФИГ. 15A-15G, где «*» обозначает любую аминокислоту, присутствующую в соответствующем положении в аминокислотной последовательности молекулы Cas9 S. mutans (SEQ ID NO: 1331); S. pyogenes (SEQ ID NO: 1332); S. thermophilus (SEQ ID NO: 1333); L. innocua (SEQ ID NO: 1334), а «-» обозначает любую аминокислоту. В одном варианте осуществления молекула Cas9 или полипептид Cas9 отличаются от последовательности консенсусной последовательности, раскрытой на ФИГ. 15A-15G, по меньшей мере, на 1, но не более чем на 2, 3, 4, 5, 6, 7, 8, 9 или 10 аминокислотных остатков. В одном варианте осуществления молекула Cas9 или полипептид Cas9 содержат аминокислотную последовательность SEQ ID NO: 1336 ФИГ. 16A-16C, где «*» обозначает любую аминокислоту, присутствующую в соответствующем положении в аминокислотной последовательности молекулы Cas9 S. pyogenes (SEQ ID NO: 1336) или N. meningitides (SEQ ID NO: 1335), «-» обозначает любую аминокислоту или ее отсутствие. В одном варианте осуществления молекула Cas9 или полипептид Cas9 отличаются от последовательности SEQ ID NO: 1335 или 1336, раскрытой на ФИГ. 16A-16C по меньшей мере на 1, но не более чем на 2, 3, 4, 5, 6, 7, 8, 9 или 10 аминокислотных остатков.
[0711] Сравнение последовательности ряда молекул Cas9 показывает, что определенные области являются консервативными. Они идентифицированы ниже как:
область 1 (остатки от 1 до 180, или в случае области 1' остатки от 120 до 180)
область 2 (остатки от 360 до 480);
область 3 (остатки от 660 до 720);
область 4 (остатки от 817 до 900); и
область 5 (остатки от 900 до 960);
[0712] В одном варианте осуществления молекула Cas9 или полипептид Cas9 содержат области 1-5 вместе с достаточной дополнительной последовательностью молекулы Cas9 для обеспечения биологически активной молекулы, например молекулы Cas9, обладающей по меньшей мере одной активностью, описанной в настоящем документе. В одном варианте осуществления каждая из областей 1-6 независимо имеет 50%, 60%, 70% или 80% гомологии с соответствующими остатками молекулы Cas9 или полипептида Cas9, описанными в настоящем документе, например, последовательностью из ФИГ. 15A-15G или из ФИГ. 16A-16C.
[0713] В одном варианте осуществления молекула Cas9 или полипептид Cas9, например молекула eaCas9 или полипептид eaCas9, содержат аминокислотную последовательность, обозначенную как область 1: имеющую 50%, 60%, 70%, 80%, 85%, 90%, 95%, 96%, 97%, 98% или 99% гомологии с аминокислотами 1-180 (нумерация соответствует последовательности мотивов на ФИГ. 15A-15G; 52% остатков в четырех последовательностях Cas9 на ФИГ. 15A-15G консервативны) аминокислотной последовательности Cas9 S. pyogenes; которая отличается по меньшей мере на 1, 2, 5, 10 или 20 аминокислот, но не более чем на 90, 80, 70, 60, 50, 40 или 30 аминокислот от аминокислот 1-180 аминокислотной последовательности Cas9 S. pyogenes, S. thermophilus, S. mutans или L. innocua; или которая идентична аминокислотам 1-180 аминокислотной последовательности Cas9 S. pyogenes, S. thermophilus, S. mutans или L. innocua.
[0714] В одном варианте осуществления молекула Cas9 или полипептид Cas9, например молекула eaCas9 или полипептид eaCas9, содержат аминокислотную последовательность, обозначенную как область 1': имеющую 55%, 60%, 65%, 70%, 75%, 80% 85%, 90%, 95%, 96%, 97%, 98% или 99% гомологии с аминокислотами 120-180 (55% остатков в четырех последовательностях Cas9 на ФИГ. 15A-15G консервативны) аминокислотной последовательности Cas9 S. pyogenes, S. thermophilus, S. mutans или L. innocua; которая отличается по меньшей мере на 1, 2 или 5 аминокислот, но не более чем на 35, 30, 25, 20 или 10 аминокислот от аминокислот 120-180 аминокислотной последовательности Cas9 S. pyogenes, S. thermophilus, S. mutans или L. innocua; или которая идентична аминокислотам 120-180 аминокислотной последовательности Cas9 S. pyogenes, S. thermophilus, S. mutans или L. innocua.
[0715] В одном варианте осуществления молекула Cas9 или полипептид Cas9, например молекула eaCas9 или полипептид eaCas9, содержат аминокислотную последовательность, обозначенную как область 2: имеющую 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98% или 99% гомологии с аминокислотами 360-480 (52% остатков в четырех последовательностях Cas9 на ФИГ. 11A-11G консервативны) аминокислотной последовательности Cas9 S. pyogenes, S. thermophilus, S. mutans или L. innocua; которая отличается по меньшей мере на 1, 2 или 5 аминокислот, но не более чем на 35, 30 25, 20 или 10 аминокислот от аминокислот 360-480 аминокислотной последовательности Cas9 S. pyogenes, S. thermophilus, S. mutans или L. innocua; или которая идентична аминокислоам 360-480 аминокислотной последовательности Cas9 S. pyogenes, S. thermophilus, S. mutans или L. innocua.
[0716] В одном варианте осуществления молекула Cas9 или полипептид Cas9, например молекула eaCas9 или полипептид eaCas9, содержат аминокислотную последовательность, обозначенную как область 3: имеющую 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98% или 99% гомологии с аминокислотами 660-720 (56% остатков в четырех последовательностях Cas9 на ФИГ. 15A-15G консервативны) аминокислотной последовательности Cas9 S. pyogenes, S. thermophilus, S. mutans или L. innocua; которая отличается по меньшей мере на 1, 2 или 5 аминокислот, но не более чем на 35, 30, 25, 20 или 10 аминокислот от аминокислот 660-720 аминокислотной последовательности Cas9 S. pyogenes, S. thermophilus, S. mutans или L. innocua; или которая идентична аминокислоам 660-720 аминокислотной последовательности Cas9 S. pyogenes, S. thermophilus, S. mutans или L. innocua.
[0717] В одном варианте осуществления молекула Cas9 или полипептид Cas9, например молекула eaCas9 или полипептид eaCas9, содержат аминокислотную последовательность, обозначенную как область 4: имеющую 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98% или 99% гомологии с аминокислотами 817-900 (55% остатков в четырех последовательностях Cas9 на ФИГ. 11A-11G консервативны) аминокислотной последовательности Cas9 S. pyogenes, S. thermophilus, S. mutans или L. innocua; которая отличается по меньшей мере на 1, 2 или 5 аминокислот, но не более чем на 35, 30, 25, 20 или 10 аминокислот от аминокислот 817-900 аминокислотной последовательности Cas9 S. pyogenes, S. thermophilus, S. mutans или L. innocua; или которая идентична аминокислотам 817-900 аминокислотной последовательности Cas9 S. pyogenes, S. thermophilus, S. mutans или L. innocua.
[0718] В одном варианте осуществления молекула Cas9 или полипептид Cas9, например молекула eaCas9 или полипептид eaCas9, содержат аминокислотную последовательность, обозначенную как область 5: имеющую 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98% или 99% гомологии с аминокислотами 900-960 (60% остатков в четырех последовательностях Cas9 на ФИГ. 15A-15G консервативны) аминокислотной последовательности Cas9 S. pyogenes, S. thermophilus, S. mutans или L. innocua; которая отличается по меньшей мере на 1, 2 или 5 аминокислот, но не более чем на 35, 30, 25, 20 или 10 аминокислот от аминокислот 900-960 аминокислотной последовательности Cas9 S. pyogenes, S. thermophilus, S. mutans или L. innocua; или которая идентична аминокислотам 900-960 аминокислотной последовательности Cas9 S. pyogenes, S. thermophilus, S. mutans или L. innocua.
(3) Сконструированные или измененные молекулы Cas9 и полипептиды Cas9
[0719] Молекулы Cas9 и полипептиды Cas9, описанные в настоящем документе, например встречающиеся в природе молекулы Cas9, могут обладать любым из ряда свойств, включая: активность никазы, активность нуклеазы (например, активность эндонуклеазы и/или экзонуклеазы); хеликазнаяую активность; способность функционально связываться с молекулой гРНК; и способность нацеливаться (или локализоваться) на сайт на нуклеиновой кислоте (например, распознавание и специфичность PAM). В одном варианте осуществления молекула Cas9 или полипептид Cas9 могут включать все или часть этих свойств. В иллюстративных вариантах осуществления молекула Cas9 или полипептид Cas9 обладают способностью взаимодействовать с молекулой гРНК и совместно с молекулой гРНК локализоваться в сайте на нуклеиновой кислоте. Другие активности, например, специфичность PAM, активность расщепления или хеликазная активность, могут варьироваться более широко в молекулах Cas9 и полипептидах Cas9.
[0720] Молекулы Cas9 включают сконструированные молекулы Cas9 и сконструированные полипептиды Cas9 («сконструированный», при использовании в данном контексте, означает лишь то, что молекула Cas9 или полипептид Cas9 отличаются от эталонных последовательностей и не предполагают никаких ограничений по способу или их происхождению). Сконструированная молекула Cas9 или полипептид Cas9 могут обладать измененными ферментативными свойствами, например, измененной нуклеазной активностью (по сравнению с природной или другой эталонной молекулой Cas9) или измененной хеликазной активностью. Как обсуждалось в настоящем документе, сконструированная молекула Cas9 или полипептид Cas9 могут обладать никазной активностью (в отличие от двухцепочечной нуклеазной активности). В одном варианте осуществления сконструированная молекула Cas9 или полипептид Cas9 могут иметь изменение, которое изменяет их размер, например делецию аминокислотной последовательности, которая уменьшает ее размер, например, без значительного влияния на одну или более или на любую активность Cas9. В одном варианте осуществления сконструированная молекула Cas9 или полипептид Cas9 могут иметь изменение, которое влияет на распознавание PAM. Например, сконструированная молекула Cas9 может быть изменена для распознавания последовательности PAM, отличной от той, которая распознается эндогенным PI-доменом дикого типа. В одном варианте осуществления молекула Cas9 или полипептид Cas9 могут отличаться по последовательности от встречающейся в природе молекулы Cas9, но не имеют пр этом значительного изменения в одной или более активностях Cas9.
[0721] Молекулы Cas9 или полипептиды Cas9 с желаемыми свойствами могут быть получены различными способами, например, путем изменения родительских, например, встречающихся в природе молекул Cas9 или полипептидов Cas9, для получения измененной молекулы Cas9 или полипептида Cas9, обладающих желаемым свойством. Например, может быть введена одна или более мутаций или различий относительно родительской молекулы Cas9, например, природной или сконструированной молекулы Cas9. Такие мутации и различия включают: замены (например, консервативные замены или замены несущественных аминокислот); вставки; или делеции. В одном варианте осуществления молекула Cas9 или полипептид Cas9 может содержать одну или более мутаций или различий, например, по меньшей мере 1, 2, 3, 4, 5, 10, 15, 20, 30, 40 или 50 мутаций, но менее чем 200, 100 или 80 мутаций относительно эталонной, например, родительской молекулы Cas9.
[0722] В одном варианте осуществления мутация или мутации не оказывают существенного влияния на активность Cas9, например активность Cas9, описанной в настоящем документе. В одном варианте осуществления мутация или мутации оказывают существенное влияние на активность Cas9, например активность Cas9, описанную в настоящем документе.
(a) Не расщепляющие молекулы и полипептиды Cas9 и обладающие свойствами модифицированного расщепления
[0723] В одном варианте осуществления молекула Cas9 или полипептид Cas9 обладают свойством расщепления, которое отличается от встречающихся в природе молекул Cas9, например, которое отличается от встречающейся в природе молекулы Cas9, имеющей наиболее близкую гомологию. Например, молекула Cas9 или полипептид Cas9 может отличаться от встречающихся в природе молекул Cas9, например молекулы Cas9 S. pyogenes, следующим образом: ее способностью модулировать, например, уменьшать или увеличивать расщепление двухцепочечной нуклеиновой кислоты (эндонуклеазная и/или экзонуклеазная активность), например, по сравнению с природной молекулой Cas9 (например, молекулой Cas9 S. pyogenes); ее способностью модулировать, например, уменьшать или увеличивать расщепление одной цепи нуклеиновой кислоты, например некомплементарной цепи молекулы нуклеиновой кислоты или комплементарной цепи молекулы нуклеиновой кислоты (активность никазы), например, по сравнению с природной молекулой Cas9 (например, молекулой Cas9 S. pyogenes); или способность расщеплять молекулу нуклеиновой кислоты, например, двухцепочечной или одноцепочечной молекулы нуклеиновой кислоты, может быть устранена.
(b) Модифицированное расщепление молекул eaCas9 и полипептидов eaCas9
[0724] В одном варианте осуществления молекула eaCas9 или полипептид eaCas9 включает одну или более из следующих активностей: активность расщепления, ассоциированную с N-концевым RuvC-подобным доменом; активность расщепления, ассоциированную с HNH-подобным доменом; активность расщепления, ассоциированную с HNH-подобным доменом, и активность расщепления, ассоциированную с N-концевым RuvC-подобным доменом.
[0725] В одном варианте осуществления молекула eaCas9 или полипептид eaCas9 содержат активный или способный к расщеплению HNH-подобный домен и неактивный или неспособный к расщеплению N-концевой RuvC-подобный домен. Иллюстративный неактивный или неспособный к расщеплению N-концевой RuvC-подобный домен может иметь мутацию аспарагиновой кислоты в N-концевом RuvC-подобном домене, например аспарагиновой кислоты в положении 9 консенсусной последовательности, раскрытой на ФИГ. 15A-15G, или аспарагиновая кислота в положении 10 SEQ ID NO: 1336, например, может быть заменена аланином. В одном варианте осуществления молекула eaCas9 или полипептид eaCas9 отличаются от дикого типа в N-концевом RuvC-подобном домене и не расщепляют нуклеиновую кислоту-мишень или расщепляют со значительно меньшей эффективностью, например с эффективностью менее чем 20, 10, 5, 1 или 0,1% активности расщепления эталонной молекулы Cas9, например, как измерено анализом, описанным в настоящем документе. Эталонная молекула Cas9 может быть природной немодифицированной молекулой Cas9, например, природной молекулой Cas9, такой как молекула Cas9 S. pyogenes или S. thermophilus. В одном варианте осуществления эталонная молекула Cas9 представляет собой природную молекулу Cas9, имеющую наиболее близкую идентичность или гомологию последовательности.
[0726] В одном варианте осуществления молекула eaCas9 или полипептид eaCas9 содержат неактивный или неспособный к расщеплению домен HNH и активный или способный к расщеплению N-концевой RuvC-подобный домен. Иллюстративные неактивные или неспособные к расщеплению HNH-подобные домены могут иметь мутацию в одном или более из: гистидина в HNH-подобном домене, например гистидин, представленный в положении 856 на ФИГ. 15A-15G, например, может быть заменен аланином; и один или более аспарагинов в HNH-подобном домене, например аспарагин, представленный в положении 870 на ФИГ. 15A-15G и/или в положении 879 на ФИГ. 15A-15G, например, может быть заменен аланином. В одном варианте осуществления eaCas9 отличается от дикого типа в HNH-подобном домене и не расщепляет нуклеиновую кислоту-мишень, или расщепляет со значительно меньшей эффективностью, например, с эффективностью менее чем 20, 10, 5, 1 или 0,1% от активности расщепления эталонной молекулы Cas9, например, как измерено анализом, описанным в настоящем документе. Эталонная молекула Cas9 может быть природной немодифицированной молекулой Cas9, например, природной молекулой Cas9, такой как молекула Cas9 S. pyogenes или S. thermophilus. В одном варианте осуществления эталонная молекула Cas9 представляет собой природную молекулу Cas9, имеющую наиболее близкую идентичность или гомологию последовательности.
[0727] В одном варианте осуществления молекула eaCas9 или полипептид eaCas9 содержат неактивный или неспособный к расщеплению домен HNH и активный или способный к расщеплению N-концевой RuvC-подобный домен. Иллюстративные неактивные или неспособные к расщеплению HNH-подобные домены могут иметь мутацию в одном или более из: гистидина в HNH-подобном домене, например гистидин, представленный в положении 856 на ФИГ. 15A-15G, например, может быть заменен аланином; и один или более аспарагинов в HNH-подобном домене, например аспарагин, представленный в положении 870 на ФИГ. 15A-15G и/или в положении 879 на ФИГ. 15A-15G, например, может быть заменен аланином. В одном варианте осуществления eaCas9 отличается от дикого типа в HNH-подобном домене и не расщепляет нуклеиновую кислоту-мишень, или расщепляет со значительно меньшей эффективностью, например, с эффективностью менее чем 20, 10, 5, 1 или 0,1% от активности расщепления эталонной молекулы Cas9, например, как измерено анализом, описанным в настоящем документе. Эталонная молекула Cas9 может быть природной немодифицированной молекулой Cas9, например, природной молекулой Cas9, такой как молекула Cas9 S. pyogenes или S. thermophilus. В одном варианте осуществления эталонная молекула Cas9 представляет собой природную молекулу Cas9, имеющую наиболее близкую идентичность или гомологию последовательности.
(c) Изменения в способности расщеплять одну или обе цепи нуклеиновой кислоты-мишени
[0728] В одном варианте осуществления иллюстративные активности Cas9 включают одну или более из специфичности PAM, активности расщепления и хеликазной активности. Мутация (мутации) может присутствовать, например, в: одном или более RuvC-подобных доменах, например, N-концевом RuvC-подобном домене; HNH-подобном домене; в области вне RuvC-подобных доменов и HNH-подобного домена. В некоторых вариантах осуществления мутация (мутации) присутствует в RuvC-подобном домене, например, в N-концевом RuvC-подобном домене. В некоторых вариантах осуществления мутация (мутации) присутствует в HNH-подобном домене. В некоторых вариантах осуществления мутации присутствуют как в RuvC-подобном домене, например, в N-концевом RuvC-подобном домене, так и в HNH-подобном домене.
[0729] Иллюстративные мутации, которые могут быть сделаны в домене RuvC или домене HNH относительно последовательности S. pyogenes, включают: D10A, E762A, H840A, N854A, N863A и/или D986A.
[0730] В одном варианте осуществления молекула Cas9 или полипептид Cas9 представляют собой молекулу eiCas9 или полипептид eiCas9, имеющие одно или более различий в домене RuvC и/или в домене HNH по сравнению с эталонной молекулой Cas9, и молекула eiCas9 или полипептид eiCas9 не расщепляют нуклеиновую кислоту или расщепляют со значительно меньшей эффективностью, чем дикий тип, например, по сравнению с диким типом в анализе расщепления, например, как описано в настоящем документе, расщепляют с эффективностью менее чем 50, 25, 10 или 1% от эталонной Cas9 молекулы, как измерено анализом, описанным в настоящем документе.
[0731] Может ли конкретная последовательность, например замена, повлиять на одну или более активностей, таких как активность нацеливания, активность расщепления и т. д., можно оценить или предсказать, например, путем оценки того, является ли мутация консервативной, или с помощью способа, описанного в Разделе IV. В одном варианте осуществления «несущественный» аминокислотный остаток, используемый в контексте молекулы Cas9, представляет собой остаток, который может быть изменен в последовательности дикого типа молекулы Cas9, например, встречающейся в природе молекулы Cas9, например, молекулы eaCas9, без аннулирования или, более предпочтительно, без существенного изменения активности Cas9 (например, активности расщепления), тогда как изменение «незаменимого» аминокислотного остатка приводит к значительной потере активности (например, активности расщепления).
[0732] В одном варианте осуществления молекула Cas9 или полипептид Cas9 обладают свойством расщепления, которое отличается от встречающихся в природе молекул Cas9, например, которое отличается от встречающейся в природе молекулы Cas9, имеющей наиболее близкую гомологию. Например, молекула Cas9 или полипептид Cas9 могут отличаться от встречающихся в природе молекул Cas9, например молекулы Cas9 S aureus, S. pyogenes или C. jejuni, следующим образом: их способностью модулировать, например, уменьшать или увеличивать расщепление двухцепочечного разрыва (эндонуклеазная и/или экзонуклеазная активность), например, по сравнению с природной молекулой Cas9 (например, молекулой Cas9 S aureus, S. pyogenes или C. jejuni); их способностью модулировать, например, уменьшать или увеличивать расщепление одной цепи нуклеиновой кислоты, например некомплементарной цепи молекулы нуклеиновой кислоты или комплементарной цепи молекулы нуклеиновой кислоты (активность никазы), например, как по сравнению с природной молекулой Cas9 (например, молекулой Cas9 S aureus, S. pyogenes или C. jejuni); или способность расщеплять молекулу нуклеиновой кислоты, например, двухцепочечную или одноцепочечную молекулу нуклеиновой кислоты, может быть устранена.
[0733] В одном варианте осуществления измененные молекула Cas9 или полипептид Cas9 представляют собой молекулу eaCas9 или полипептид eaCas9, обладающие одной или более из следующих активностей: активностью расщепления, ассоциированной с доменом RuvC; активностью расщепления, ассоциированной с доменом HNH; активностью расщепления, ассоциированной с доменом HNH, и активностью расщепления, ассоциированной с доменом RuvC.
[0734] В одном варианте осуществления измененные молекула Cas9 или полипептид Cas9 представляют собой молекулу eiCas9 или полипептид eaCas9, которые не расщепляют молекулу нуклеиновой кислоты (ни двухцепочечную, ни одноцепочечную молекулы нуклеиновой кислоты) или расщепляют молекулу нуклеиновой кислоты со значительно меньшей эффективностью, например, с эффективностью менее чем 20, 10, 5, 1 или 0,1% от активности расщепления эталонной молекулы Cas9, например, как измерено анализом, описанным в настоящем документе. Эталонная молекула Cas9 может быть природной немодифицированной молекулой Cas9, например, природной молекулой Cas9, такой как молекула Cas9 S. pyogenes, S. thermophilus, S. aureus, C. jejuni или N. meningitidis. В одном варианте осуществления эталонная молекула Cas9 является природной молекулой Cas9, имеющей наиболее близкую идентичность или гомологию последовательности. В одном варианте осуществления молекула eiCas9 или полипептид eiCas9 не обладают значительной активностью расщепления, ассоциированной с доменом RuvC, и активностью расщепления, ассоциированной с доменом HNH.
[0735] В одном варианте осуществления измененная молекула Cas9 или полипептид Cas9 представляют собой молекулу eaCas9 или полипептид eaCas9, содержащие фиксированные аминокислотные остатки S. pyogenes, представленные в консенсусной последовательности, раскрытой на ФИГ. 15A-15G, и имеют одну или более аминокислот, которые отличаются от аминокислотной последовательности S. pyogenes (например, имеют замену) в одном или более остатках (например, 2, 3, 5, 10, 15, 20, 30, 50, 70, 80, 90, 100, 200 аминокислотных остатков), обозначенных «-» в консенсусной последовательности, раскрытой на ФИГ. 15A-15G или в SEQ ID NO: 1336.
[0736] В одном варианте осуществления измененная молекула Cas9 или полипептид Cas9 содержат последовательность, в которой:
[0737] последовательность, соответствующая фиксированной последовательности консенсусной последовательности, раскрытой на ФИГ. 15A-15G, отличается не более чем на 1, 2, 3, 4, 5, 10, 15 или 20% фиксированных остатков в консенсусной последовательности, раскрытой на ФИГ. 15A-15G;
[0738] последовательность, соответствующая остаткам, обозначенным «*» в консенсусной последовательности, раскрытой на ФИГ. 15A-15G, отличается не более чем на 1, 2, 3, 4, 5, 10, 15, 20, 25, 30, 35 или 40% остатков «*» из соответствующей последовательности встречающейся в природе молекулы Cas9, например, молекулы Cas9 S. pyogenes; и,
[0739] последовательность, соответствующая остаткам, обозначенным «-» в консенсусной последовательности, раскрытой на ФИГ. 15A-15G, отличается не более чем на 5, 10, 15, 20, 25, 30, 35, 40, 45, 55 или 60% остатков «-» из соответствующей последовательности встречающейся в природе молекулы Cas9, например молекулы Cas9 S. Pyogenes.
[0740] В одном варианте осуществления измененная молекула Cas9 или полипептид Cas9 представляет собой молекулу eaCas9 или полипептид eaCas9, содержащие фиксированные аминокислотные остатки S. thermophilus, представленные в консенсусной последовательности, раскрытой на ФИГ. 15A-15G, и имеют одну или более аминокислот, которые отличаются от аминокислотной последовательности S. thermophilus (например, имеют замену) в одном или более остатках (например, 2, 3, 5, 10, 15, 20, 30). 50, 70, 80, 90, 100, 200 аминокислотных остатков), обозначенных «-» в консенсусной последовательности, раскрытой на ФИГ. 15А-15G.
[0741] В одном варианте осуществления измененная молекула Cas9 или полипептид Cas9 содержат последовательность, в которой:
[0742] последовательность, соответствующая фиксированной последовательности консенсусной последовательности, раскрытой на ФИГ. 15A-15G, отличается не более чем на 1, 2, 3, 4, 5, 10, 15 или 20% фиксированных остатков в консенсусной последовательности, раскрытой на ФИГ. 15A-15G;
[0743] последовательность, соответствующая остаткам, обозначенным «*» в консенсусной последовательности, раскрытой на ФИГ. 15A-15G, отличается не более чем на 1, 2, 3, 4, 5, 10, 15, 20, 25, 30, 35 или 40% остатков «*» из соответствующей последовательности встречающейся в природе молекулы Cas9, например, молекулы Cas9 S. thermophilus; и,
[0744] последовательность, соответствующая остаткам, обозначенным «-» в консенсусной последовательности, раскрытой на ФИГ. 15A-15G, отличается не более чем на 5, 10, 15, 20, 25, 30, 35, 40, 45, 55 или 60% остатков «-» из соответствующей последовательности встречающейся в природе молекулы Cas9, например молекулы Cas9 S. thermophilus.
[0745] В одном варианте осуществления измененная молекула Cas9 или полипептид Cas9 представляют собой молекулу eaCas9 или полипептид eaCas9, содержащие фиксированные аминокислотные остатки S. mutans, представленные в консенсусной последовательности, раскрытой на ФИГ. 15A-15G, и имеют одну или более аминокислот, которые отличаются от аминокислотной последовательности S. mutans (например, имеют замену) в одном или более остатках (например, в 2, 3, 5, 10, 15, 20, 30, 50, 70, 80, 90, 100, 200 аминокислотных остатках), обозначенных «-» в консенсусной последовательности, раскрытой на ФИГ. 15А-15G.
[0746] В одном варианте осуществления измененная молекула Cas9 или полипептид Cas9 содержат последовательность, в которой:
[0747] последовательность, соответствующая фиксированной последовательности консенсусной последовательности, раскрытой на ФИГ. 15A-15G, отличается не более чем на 1, 2, 3, 4, 5, 10, 15 или 20% фиксированных остатков в консенсусной последовательности, раскрытой на ФИГ. 15A-15G;
[0748] последовательность, соответствующая остаткам, обозначенным «*» в консенсусной последовательности, раскрытой на ФИГ. 15A-15G, отличается не более чем на 1, 2, 3, 4, 5, 10, 15, 20, 25, 30, 35 или 40% остатков «*» из соответствующей последовательности встречающейся в природе молекулы Cas9, например, молекулы Cas9 S. mutans; и,
[0749] последовательность, соответствующая остаткам, обозначенным «-» в консенсусной последовательности, раскрытой на ФИГ. 15A-15G, отличается не более чем на 5, 10, 15, 20, 25, 30, 35, 40, 45, 55 или 60% остатков «-» из соответствующей последовательности встречающейся в природе молекулы Cas9, например молекулы Cas9 S. mutans.
[0750] В одном варианте осуществления измененная молекула Cas9 или полипептид Cas9 представляют собой молекулу eaCas9 или полипептид eaCas9, содержащие фиксированные аминокислотные остатки L. innocula, представленные в консенсусной последовательности, раскрытой на ФИГ. 15A-15G, и имеет одну или более аминокислот, которые отличаются от аминокислотной последовательности L. innocula (например, имеют замену) в одном или более остатках (например, в 2, 3, 5, 10, 15, 20, 30, 50, 70, 80, 90, 100, 200 аминокислотных остатках), обозначенных «-» в консенсусной последовательности, раскрытой на ФИГ. 15А-15G.
[0751] В одном варианте осуществления измененная молекула Cas9 или полипептид Cas9 содержат последовательность, в которой:
[0752] последовательность, соответствующая фиксированной последовательности консенсусной последовательности, раскрытой на ФИГ. 15A-15G, отличается не более чем на 1, 2, 3, 4, 5, 10, 15 или 20% фиксированных остатков в консенсусной последовательности, раскрытой на ФИГ. 15A-15G;
[0753] последовательность, соответствующая остаткам, обозначенным «*» в консенсусной последовательности, раскрытой на ФИГ. 15A-15G, отличаются не более чем на 1, 2, 3, 4, 5, 10, 15, 20, 25, 30, 35 или 40% остатков «*» из соответствующей последовательности встречающейся в природе молекулы Cas9, например, молекулы Cas9 L. innocula; и,
[0754] последовательность, соответствующая остаткам, обозначенным «-» в консенсусной последовательности, раскрытой на ФИГ. 15A-15G, отличаются не более чем на 5, 10, 15, 20, 25, 30, 35, 40, 45, 55 или 60% остатков «-» из соответствующей последовательности встречающейся в природе молекулы Cas9, например молекулы Cas9 L. Innocula.
[0755] В одном варианте осуществления измененная молекула Cas9 или полипептид Cas9, например молекула eaCas9, могут быть слиянием, например, двух или более различных молекул Cas9 или полипептидов Cas9, например, двух или более встречающихся в природе молекул Cas9 разных видов. Например, фрагмент встречающейся в природе молекулы Cas9 одного вида может быть слит с фрагментом молекулы Cas9 второго вида. Например, фрагмент молекулы Cas9 S. pyogenes, содержащий N-концевой RuvC-подобный домен, может быть слит с фрагментом молекулы Cas9 вида, отличного от S. pyogenes (например, S. thermophilus), содержащего HNH-подобный домен.
(d) Молекулы Cas9 с измененным распознаванием PAM или без распознавания PAM
[0756] Встречающиеся в природе молекулы Cas9 могут распознавать конкретные последовательности PAM, например, последовательности распознавания PAM, описанные выше, например, для S. pyogenes, S. thermophilus, S. mutans, S. aureus и N. meningitidis.
[0757] В одном варианте осуществления молекула Cas9 или полипептид Cas9 обладают такими же специфичностями PAM, что и встречающаяся в природе молекула Cas9. В других вариантах осуществления молекула Cas9 или полипептид Cas9 имеет специфичность PAM, не связанную с природной молекулой Cas9, или специфичность PAM, не связанную с природной молекулой Cas9, с которой она имеет наиболее близкую гомологию последовательности. Например, встречающаяся в природе молекула Cas9 может быть изменена, например, для изменения распознавания PAM, например, для изменения последовательности PAM, которую распознает молекула Cas9 или полипептид Cas9, чтобы уменьшить ошибочные сайты и/или улучшить специфичность; или исключить условия распознавания PAM. В одном варианте осуществления молекула Cas9 может быть изменена, например, для увеличения длины последовательности распознавания PAM и/или улучшения специфичности Cas9 до высокого уровня идентичности, например, для уменьшения ошибочных сайтов и увеличения специфичности. В одном варианте осуществления длина последовательности распознавания PAM составляет по меньшей мере 4, 5, 6, 7, 8, 9, 10 или 15 аминокислот.
[0758] Молекулы Cas9 или полипептиды Cas9, которые распознают различные последовательности PAM и/или имеют пониженную ошибочную активность, могут быть получены с использованием направленной эволюции. Иллюстративные способы и системы, которые можно использовать для направленной эволюции молекул Cas9, описаны, например, в Esvelt et al. Nature 2011, 472 (7344): 499-503. Молекулы-кандидаты Cas9 могут быть подвергнуты оценке.
(4) Нуклеиновые кислоты, кодирующие молекулы Cas9
[0759] В настоящем описании представлены нуклеиновые кислоты, кодирующие молекулы Cas9 или полипептиды Cas9, например молекулу eaCas9 или полипептид eaCas9.
[0760] Иллюстративные нуклеиновые кислоты, кодирующие молекулы Cas9 или полипептиды Cas9, описаны в Cong et al., Science 2013, 399 (6121): 819-823; Wang et al., Cell 2013, 153 (4): 910-918; Mali et al., Science 2013, 399 (6121): 823-826; Jinek et al., Science 2012, 337 (6096): 816-821. Другая иллюстративная нуклеиновая кислота, кодирующая молекулу Cas9 или полипептид Cas9, показана черным на ФИГ. 8 WO2015161276.
[0761] В одном варианте осуществления нуклеиновая кислота, кодирующая молекулу Cas9 или полипептид Cas9, может представлять собой синтетическую последовательность нуклеиновой кислоты. Например, синтетическая молекула нуклеиновой кислоты может быть химически модифицирована. В одном варианте осуществления мРНК Cas9 обладает одним или более (например, всеми из следующих свойств: она кэппирована, полиаденилирована, замещена 5-метилцитидином и/или псевдоуридином.
[0762] Кроме того, или альтернативно, последовательность синтетической нуклеиновой кислоты может быть оптимизирована по кодонам, например, по меньшей мере один редкий кодон или менее распространенный кодон был заменен обычным кодоном. Например, синтетическая нуклеиновая кислота может направлять синтез оптимизированной матричной мРНК, например, оптимизированной для экспрессии в системе экспрессии млекопитающего, например, описанной в настоящем документе.
[0763] Кроме того, или альтернативно, нуклеиновая кислота, кодирующая молекулу Cas9 или полипептид Cas9, может содержать последовательность ядерной локализации (NLS). Последовательности ядерной локализации известны в данной области.
[0764] SEQ ID NO: 1337 представляет собой пример оптимизированной по кодонам последовательности нуклеиновой кислоты, кодирующей молекулу Cas9 S. pyogenes. SEQ ID NO: 1338 представляет собой соответствующую аминокислотную последовательность молекулы Cas9 S. pyogenes. SEQ ID NO: 1339 представляет собой пример оптимизированной по кодонам последовательности нуклеиновой кислоты, кодирующей молекулу Cas9 N. meningitidis. SEQ ID NO: 1340 представляет собой соответствующую аминокислотную последовательность молекулы Cas9 N. meningitidis. SEQ ID NO: 1341 представляет собой пример оптимизированной по кодонам последовательности нуклеиновой кислоты, кодирующей молекулу Cas9 S. aureus. SEQ ID NO: 1342 представляет собой соответствующую аминокислотную последовательность молекулы Cas9 S. aureus.
[0765] Если какая-либо из указанных выше последовательностей Cas9 слита с пептидом или полипептидом на С-конце, подразумевается, что стоп-кодон будет удален.
(5) Другие молекулы Cas и полипептиды Cas
[0766] Различные типы молекул Cas или полипептидов Cas могут быть использованы для практического использования раскрытого в настоящем описании изобретения. В некоторых вариантах осуществления используются молекулы Cas систем Cas типа II. В других вариантах осуществления используются молекулы Cas других систем Cas. Например, могут использоваться молекулы Cas типа I или типа III. Примеры молекул Cas (и систем Cas) описаны, например, в Haft et al., PLoS Computational Biology 2005, 1 (6): e60 и Makarova et al., Nature Review Microbiology 2011, 9: 467-477, содержание обеих ссылок включено в настоящее описание в качестве ссылки в полном объеме. Примеры молекул Cas (и систем Cas) также представлены в Таблице 18.
Таблица 18 Системы Cas
Тип II
Тип III
Тип II
Тип III
• Подтип I-B
• Подтип I-B
• Подтип I-C
• Подтип I-D
• Подтип II-B
• Подтип I-B
•Подтип I-C
• Подтип I-E
•Подтип I-B
•Подтип I-D
•Подтип III-A
• Подтип III-B
• Подтип I-B
• Подтип I-C
• Подтип I-E
c) Cpf1
[0767] В некоторых вариантах осуществления гид-РНК или гРНК стимулирует специфическое нацеливание ассоциации РНК-управляемой нуклеазы, такой как Cas9 или Cpf1, на последовательность-мишень, такую как геномная или эписомная последовательность в клетке. В целом, гРНК могут быть мономолекулярными (содержащими одну молекулу РНК и альтернативно называются химерными) или модульными (содержащими более чем одну и обычно две отдельные молекулы РНК, такие как crРНК и tracrРНК, которые обычно связаны друг с другом, например, путем образования дуплекса). гРНК и их составные части описаны в литературе, например, в Briner et al. (Molecular Cell 56(2), 333-339, October 23, 2014 (Briner), которая включена в качестве ссылки), и у Cotta-Ramusino.
[0768] гид-РНК, будь то мономолекулярные или модульные, обычно включают нацеливающий домен, который полностью или частично комплементарен мишени, и обычно имеет длину 10-30 нуклеотидов, а в некоторых вариантах осуществления длина составляет 16-24 нуклеотида (например, 16, 17, 18, 19, 20, 21, 22, 23 или 24 нуклеотида в длину). В некоторых аспектах нацеливающие домены находятся на или вблизи 5'-конца гРНК в случае с Cas9 гРНК, и на или примерно в близи 3'-конца в случае Cpf1 гРНК. Хотя вышеприведенное описание было сфокусировано на гРНК для использования с Cas9, следует понимать, что были обнаружены или изобретены другие РНК-направляемые нуклеазы, которые используют гРНК, которые в некоторых отношениях отличаются от описанных к настоящему моменту. Например, Cpf1 («CRISPR от Prevotella и Franciscella 1») является недавно обнаруженной РНК-управляемой нуклеазой, для функционирования которой не требуется tracrРНК. (Zetsche et al., 2015, Cell 163, 759-771, October 22, 2015 (Zetsche I), включено в настоящее описание в качестве ссылки). гРНК для использования в системе редактирования генома Cpf1 обычно включает нацеливающий домен и домен комплементарности (поочередно называемый «handle»). Следует также отметить, что в гРНК для использования с Cpf1 нацеливающий домен обычно присутствует на 3'-конце или вблизи него, а не на 5'-конце, как описано выше в связи с гРНК Cas9 («handle» находится на 5'-конце гРНК Cpf1 или вблизи него).
[0769] Хотя структурные различия могут существовать между гРНК из разных прокариотических видов или между гРНК Cpf1 и Cas9, принципы, с помощью которых действуют эти гРНК, в целом согласуются. Благодаря этой последовательности действий, гРНК могут быть определены, в широком смысле, по их последовательностям нацеливающих доменов, и квалифицированные специалисты поймут, что данная последовательность нацеливающих доменов может быть включена в любую подходящую гРНК, включая мономолекулярную или химерную гРНК, или гРНК, которая включает одну или более химических модификаций и/или последовательных модификаций (замены, дополнительные нуклеотиды, укорачивания и т. д.). Таким образом, в некоторых аспектах в настоящем раскрытии, гРНК могут быть описаны исключительно в терминах их последовательностей нацеливающих доменов.
[0770] В более общем смысле, некоторые аспекты настоящего раскрытия относятся к системам, способам и композициям, которые могут быть реализованы с использованием множества РНК-управляемых нуклеаз. Если не указано иное, термин «гРНК» следует понимать как охватывающий любую подходящую гРНК, которая может использоваться с любой РНК-управляемой нуклеазой, а не только те гРНК, которые совместимы с конкретным видом Cas9 или Cpf1. В качестве иллюстрации, термин «гРНК» может в некоторых вариантах осуществления включать в себя гРНК для использования с любой РНК-управляемой нуклеазой, встречающейся в системе CRISPR класса 2, такой как система типа II или типа V или CRISPR, или в системе с РНК-управляемой нуклеазой, полученной или адаптированной из них.
[0771] Некоторые иллюстративные модификации, обсуждаемые в этом разделе, могут быть включены в любом положении в последовательности гРНК, включая, без ограничения, на 5'-конце или вблизи него (например, в пределах 1-10, 1-5 или 1-2 нуклеотидов от 5'-конца) и/или на 3'-конце или вблизи него (например, в пределах 1-10, 1-5 или 1-2 нуклеотидов от 3'-конца). В некоторых случаях модификации располагаются в пределах функциональных мотивов, таких как дуплекс повтор-анти-повтор гРНК Cas9, структура стебель-петля гРНК Cas9 или Cpf1 и/или нацеливающий домен гРНК.
[0772] РНК-направляемые нуклеазы включают, но не ограничиваются ими, природные нуклеазы CRISPR класса 2, такие как Cas9 и Cpf1, а также другие нуклеазы, выделенные или полученные из них. С функциональной точки зрения РНК-направляемые нуклеазы определяются как нуклеазы, которые: (a) взаимодействуют (например, образуют комплекс) с гРНК; и (b) вместе с гРНК связываются и необязательно расщепляют или модифицируют область-мишень ДНК, которая включает (i) последовательность, комплементарную нацеливающему домену гРНК, и, необязательно, (ii) дополнительную последовательность, обозначенную как «мотив, смежный с протоспейсером» или «PAM», который более подробно описан ниже. Как проиллюстрированы следующие примеры, РНК-направляемые нуклеазы могут быть определены в широком смысле их специфичностью к PAM и активностью расщепления, даже если могут существовать различия между отдельными РНК-управляемыми нуклеазами, которые обладают одинаковой специфичностью к PAM или активностью расщепления. Специалистам в данной области техники будет понятно, что некоторые аспекты настоящего раскрытия относятся к системам, способам и композициям, которые могут быть реализованы с использованием любой подходящей РНК-управляемой нуклеазы, обладающей определенной специфичностью к PAM и/или активностью расщепления. По этой причине, если не указано иное, термин «РНК-управляемая нуклеаза» следует понимать как общий термин, и не ограничивается каким-либо конкретным типом (например, Cas9 по сравнению с Cpf1), видами (например, S. pyogenes по сравнению с S. aureus) или вариация (например, полная длина по сравнению с укороченной или расщепленной; прродная специфичность PAM по сравнению со сконструированной специфичностью к PAM и т. д.) РНК-управляемой нуклеазы.
[0773] В дополнение к распознаванию специфических последовательных ориентаций PAM и протоспейсеров, РНК-направляемые нуклеазы в некоторых вариантах осуществления также могут распознавать специфические последовательности PAM. Например, Cas9 S. aureus, как правило, распознает последовательность PAM NNGRRT или NNGRRV, где N остатков находятся непосредственно в 3'-области относительно области, распознаваемой нацеливающим доменом гРНК. Cas9 S. pyogenes обычно распознает последовательности NGG PAM. И Cpf1 F. novicida обычно распознает последовательность TTN PAM.
[0774] Кристаллическая структура Cpf1 Acidaminococcus sp. в комплексе с crРНК и двухцепочечной (ds) ДНК-мишенью, включающей последовательность TTTN PAM, была разрешена Yamano et al. (Cell. 2016 May 5; 165(4): 949-962 (Yamano), включено в настоящее описание в качестве ссылки). Cpf1, как и Cas9, имеет две доли: долю REC (распознавания) и долю NUC (нуклеазы). Доля REC включает в себя домены REC1 и REC2, которые не имеют сходства с какими-либо известными белковыми структурами. В то же время доля NUC включает три домена RuvC (RuvC-I, -II и -III) и домен BH. Однако, в отличие от Cas9, доля REC Cpf1 не имеет домена HNH и включает в себя другие домены, которые также не имеют сходства с известными белковыми структурами: структурно уникальный домен PI, три клиновидных домена (Wedge) (WED) (WED-I, -II и - III) и нуклеазный (Nuc) домен.
[0775] Хотя Cas9 и Cpf1 имеют сходные структуры и функции, следует понимать, что определенные активности Cpf1 опосредуются структурными доменами, которые не аналогичны каким-либо доменам Cas9. Например, расщепление комплементарной цепи ДНК-мишени, по-видимому, опосредовано доменом Nuc, который по последовательности и пространственно отличается от домена HNH в Cas9. Кроме того, не нацеливающая часть гРНК Cpf1 (handle) принимает структуру псевдоузла, а не структуру стебель-петля, образованную дуплексом повтор:антиповтр в гРНК Cas9.
[0776] Нуклеиновые кислоты, кодирующие РНК-направляемые нуклеазы, например Cas9, Cpf1 или их функциональные фрагменты, предложены в настоящем описании. Иллюстративные нуклеиновые кислоты, кодирующие РНК-направляемые нуклеазы, были описаны ранее (см., например, Cong 2013; Wang 2013; Mali 2013; Jinek 2012).
3. Методы редактирования генома и методы доставки
a) Подходы к редактированию генома
[0777] В общем, следует понимать, что изменение любого гена в соответствии со способами, описанными в настоящем документе, может быть опосредовано любым механизмом, и что любые способы не ограничиваются конкретным механизмом. Иллюстративные механизмы, которые могут быть связаны с изменением гена, включают, но не ограничиваются этим, негомологичное соединение концов (например, классическое или альтернативное), микрогомологичное соединение концов (MMEJ), направляемая гомологией репарация (например, опосредованная эндогенной донорной матрицей), синтез-зависимый отжиг цепи (SDSA), отжиг одной цепи, инвазия одной цепи, репарация одноцепочечного разрыва (SSBR), репарация ошибочного спаривания (MMR), эксцизионная репарация оснований (BER), межцепочечная сшивка (ICL), синтез ДНК «через повреждения» (TLS), или безошибочная пострепликативная репарация (PRR). В настоящем документе описаны иллюстративные способы направленного нокаута одного или обоих аллелей одного или всех TRAC, TRBC1 и/или TRBC2.
(1) Подходы NHEJ для направленного воздействия на гены
[0778] Как описано в настоящем документе, индуцированное нуклеазой негомологичное соединение концов (NHEJ) может использоваться для нацеливания на ген-специфические нокауты. Индуцированное нуклеазой NHEJ также можно использовать для удаления (например, делеции) вставок последовательности в представляющем интерес гене.
[0779] Не желая быть связанными теорией, полагают, что в одном варианте осуществления геномные изменения, связанные с описанными в настоящем документе способами, основаны на индуцированном нуклеазой NHEJ и характере пониженной точности пути репарации NHEJ. NHEJ восстанавливает двухцепочечный разрыв в ДНК, соединяя вместе два конца; однако, как правило, исходная последовательность восстанавливается только в том случае, если два совместимых конца, точно так же, как они были образованы двухцепочечным разрывом, идеально лигируются. Концы ДНК двухцепочечного разрыва часто подвергаются ферментативной обработке, приводящей к добавлению или удалению нуклеотидов в одной или обеих цепях до соединения концов. Это приводит к наличию вставочных и/или делеционных (независимых) мутаций в последовательности ДНК в месте репарации NHEJ. Две трети этих мутаций обычно изменяют рамку считывания и, следовательно, производят нефункциональный белок. Кроме того, мутации, которые поддерживают рамку считывания, но которые вставляют или удаляют значительное количество последовательности, могут разрушать функциональность белка. Это зависит от локуса, поскольку мутации в критических функциональных доменах, вероятно, менее переносимы, чем мутации в некритических областях белка. Мутации вставок-делеций, генерируемые NHEJ, непредсказуемы по своей природе; однако в данном сайте разрыва определенные последовательности вставок-делеций являются предпочтительными и чрезмерно представлены в популяции, вероятно, из-за небольших областей микрогомологии. Длина делеций может широко варьироваться; чаще всего в диапазоне 1-50 п.н., но они могут легко достигать более 100-200 п.н. Вставки, как правило, короче и часто включают короткие дубликаты последовательности, непосредственно окружающие сайт разрыва. Однако возможно получить большие вставки, и в этих случаях встроенная последовательность часто прослеживается в других областях генома или в плазмидной ДНК, присутствующей в клетках.
[0780] Поскольку NHEJ является мутагенным процессом, его также можно использовать для делеции небольших мотивов последовательности, если генерация конкретной конечной последовательности не требуется. Если двухцепочечный разрыв подвергается направленному воздействию вблизи короткой последовательности-мишени, делеционные мутации, вызванные репарацией NHEJ, часто охватывают и, следовательно, удаляют нежелательные нуклеотиды. Для удаления более крупных сегментов ДНК введение двух двухцепочечных разрывов, по одному на каждой стороне последовательности, может привести к NHEJ между концами с удалением всей промежуточной последовательности. В некоторых вариантах осуществления пара гРНК может использоваться для введения двух двухцепочечных разрывов, что приводит к удалению промежуточных последовательностей между двумя разрывами.
[0781] Оба этих подхода могут использоваться для удаления определенных последовательностей ДНК; однако характер пониженной точности NHEJ может все еще вызывать мутации в сайте репарации.
[0782] В способах и композициях, описанных в настоящем документе, для получения NHEJ-опосредованных вставок-делеций могут быть использованы как молекулы eaCas9, осуществляющие двухцепочечное расщепление, так и никазы, молекулы eaCas9, осуществляющие одноцепочечное расщепление. NHEJ-опосредованные вставки-делеции, нацеленные на ген, например кодирующую область, например, раннюю кодирующую область гена, представляющего интерес, могут быть использованы для нокаута (то есть, устранения экспрессии) представляющего интерес гена. Например, ранняя кодирующая область представляющего интерес гена включает последовательность, следующую непосредственно за стартовым сайтом транскрипции, в пределах первого экзона кодирующей последовательности или в пределах 500 п.н. от стартового сайта транскрипции (например, менее чем 500, 450, 400, 350 300, 250, 200, 150, 100 или 50 п.н.).
[0783] В одном варианте осуществления NHEJ-опосредованные вставки-делеции вводят в один или более генов, экспрессируемых T-клетками, таких как TRAC, TRBC1 и/или TRBC2. Индивидуальные гРНК или пары гРНК, нацеленные на ген, предоставляются вместе с двухцепочечной нуклеазой Cas9 или одноцепочечной никазой.
(2) Расположение двухцепочечных или одноцепочечных разрывов относительно положения-мишени
[0784] В одном варианте осуществления, в котором гРНК и нуклеаза Cas9 генерируют двухцепочечный разрыв с целью индукции NHEJ-опосредованных вставок-делеций, гРНК, например, мономолекулярная (или химерная) или модульная молекула гРНК, сконфигурирована для расположения одного двухцепочечного разрыва в непосредственной близости от нуклеотида положения-мишени. В одном варианте осуществления сайт расщепления находится на расстоянии 0-30 п.н. от положения-мишени (например, на расстоянии менее чем 30, 25, 20, 15, 10, 9, 8, 7, 6, 5, 4, 3, 2 или 1 п.н от положения-мишени).
[0785] В варианте осуществления, в котором две гРНК, образующие комплекс с никазами Cas9, индуцируют два одноцепочечных разрыва с целью индукции NHEJ-опосредованных вставок-делеций, две гРНК, например, независимо, мономолекулярная (или химерная) или модульная гРНК, сконфигурированы для расположения двух одноцепочечных разрывов, чтобы обеспечить NHEJ-репарацию нуклеотида в положении-мишени. В одном варианте осуществления, гРНК сконфигурированы для расположения разрезов в одном и том же положении или в пределах нескольких нуклеотидов друг от друга на разных цепях, по существу имитируя двухцепочечный разрыв. В одном варианте осуществления более близкий одноцепочечный разрыв находится на расстоянии 0-30 п.н. от положения-мишени (например, на расстоянии менее чем 30, 25, 20, 15, 10, 9, 8, 7, 6, 5, 4, 3, 2 или 1 п.н. от положения-мишени), и два одноцепочечных разрыва находятся в пределах 25-55 п.н. друг от друга (например, на расстоянии от 25 до 50, от 25 до 45, от 25 до 40, от 25 до 35, от 25 до 30, от 50 до 55, от 45 до 55, от 40 до 55, от 35 до 55, от 30 до 55, от 30 до 50, от 35 до 50, от 40 до 50, от 45до 50, от 35 до 45 или от 40 до 45 п.н.) и не более чем 100 п.н. друг от друга (например, не более чем 90, 80, 70, 60, 50, 40, 30, 20 или 10 п.н.). В одном варианте осуществления гРНК сконфигурированы для расположения одноцепочечного разрыва по обе стороны от нуклеотида в положении-мишени.
[0786] В способах и композициях, описанных в настоящем документе, для генерации разрывов обеих сторон положения-мишени могут быть использованы как молекулы eaCas9, осуществляющие двухцепочечное расщепление, так и никазы, молекулы eaCas9, осуществляющие одноцепочечное расщепление. Двухцепочечные или спаренные одноцепочечные разрывы могут генерироваться на обеих сторонах положения-мишени для удаления последовательности нуклеиновой кислоты между двумя разрезами (например, область между двумя разрывами делетируется). В одном варианте осуществления две гРНК, например независимо, мономолекулярная (или химерная) или модульная гРНК, сконфигурированы для расположения двухцепочечного разрыва с обеих сторон положения-мишени. В альтернативном варианте осуществления три гРНК, например, независимо, мономолекулярная (или химерная) или модульная гРНК, сконфигурированы для расположения двухцепочечного разрыва (то есть, одна гРНК образует комплекс с нуклеазой cas9) и двух одноцепочечных разрывов или спаренных одноцепочечных разрывов (т. е. две гРНК образуют комплекс с никазами Cas9) по обе стороны от положения-мишени. В другом варианте осуществления четыре гРНК, например, независимо, мономолекулярная (или химерная) или модульная гРНК, сконфигурированы для генерации двух пар одноцепочечных разрывов (то есть две пары из двух гРНК образуют комплекс с никазами Cas9) по обе стороны от положения-мишени. Двухцепочечный разрыв (разрывы) или более близкий из двух одноцепочечных разрывов в паре в идеале должен располагаться в пределах 0-500 п.н. от положения-мишени (например, на расстоянии не более чем 450, 400, 350, 300, 250, 200, 150). 100, 50 или 25 п.н. от положения-мишени). Когда используются никазы, два одноцепочечных разрыва в паре находятся в пределах 25-55 п.н. друг от друга (например, на расстоянии от 25 до 50, от 25 до 45, от 25 до 40, от 25 до 35, от 25 до 30, от 50 до 55, от 45 до 55, от 40 до 55, от 35 до 55, от 30 до 55, от 30 до 50, от 35 до 50, от 40 до 50, от 45 до 50, от 35 до 45 или от 40 до 45 п.н.) и не более чем 100 п.н. друг от друга (например, не более чем 90, 80, 70, 60, 50, 40, 30, 20 или 10 п.н.).
(3) Направленный нокдаун
[0787] В отличие от CRISPR/Cas-опосредованного нокаута гена, который надолго устраняет или уменьшает экспрессию путем мутации гена на уровне ДНК, CRISPR/Cas нокдаун позволяет временно уменьшить экспрессию гена с помощью искусственных факторов транскрипции. Мутация ключевых остатков в обоих доменах расщепления ДНК белка Cas9 (например, мутации D10A и H840A) приводит к образованию каталитически неактивного Cas9 (eiCas9, который также известен как мертвый Cas9 или dCas9). Каталитически неактивный Cas9 образует комплексы с гРНК и локализуется в последовательности ДНК, определяемой нацеливающим доменом этой гРНК, однако он не расщепляет ДНК-мишень. Слияние dCas9 с эффекторным доменом, например с доменом транскрипционной репрессии, позволяет рекрутировать эффектор в любой сайт ДНК, определяемую гРНК. Хотя было показано, что сам eiCas9 может блокировать транскрипцию при рекрутировании в ранние области в кодирующей последовательности, более надежная репрессия может быть достигнута путем слияния домена транскрипционной репрессии (например, KRAB, SID или ERD) с Cas9 и рекрутирования его в промоторную область гена. Вполне вероятно, что нацеливание ДНК-азы I на гиперчувствительные области промотора может привести к более эффективной репрессии или активации генов, поскольку эти области, более вероятно, будут доступны белку Cas9, а также с большей вероятностью содержат сайты для эндогенных факторов транскрипции. В частности, для репрессии генов здесь предусматривается, что блокирование сайта связывания эндогенного фактора транскрипции будет способствовать подавлению экспрессии гена. В другом варианте осуществления eiCas9 может быть слит с белком, модифицирующим хроматин. Изменение статуса хроматина может привести к снижению экспрессии гена-мишени.
[0788] В одном варианте осуществления молекула гРНК может быть нацелена на известные транскрипционные респонсивные элементы (например, промоторы, энхансеры и т. д.), известные последовательности активации UAS и/или последовательности неизвестной или известной функции, которые предположительно способны контролировать экспрессию ДНК-мишени.
[0789] В одном варианте осуществления CRISPR/Cas-опосредованный нокдаун гена может быть использован для снижения экспрессии одного или более генов, экспрессируемых T-клетками. В одном варианте осуществления, в котором слитый белок eiCas9 или eiCas9, описанный в настоящем документе, используется для нокдауна двух генов, экспрессируемых T-клетками, например любых двух или более генов TRAC, TRBC1 и/или TRBC2, отдельные пары гРНК или гРНК, нацеленные на оба или на все гены, обеспечиваются вместе с eiCas9 или со слитым белком eiCas9.
(4) Одноцепочечный отжиг
[0790] Рдноцепочечный отжиг (SSA) представляет собой еще один процесс репарации ДНК, который восстанавливает двухцепочечный разрыв между двумя повторяющимися последовательностями, присутствующими в нуклеиновой кислоте-мишени. Повторяющиеся последовательности, используемые SSA, обычно имеют длину более чем 30 нуклеотидов. Разрез на концах разрыва происходит для выявления повторяющихся последовательностей на обеих цепях нуклеиновой кислоты-мишени. После разреза одноцепочечные выступы, содержащие повторяющиеся последовательности, покрывают белком RPA для предотвращения несоответствующего отжига повторяющихся последовательностей, например, сами на себя. RAD52 связывается с каждой из повторяющихся последовательностей на выступающих концах и выравнивает их, чтобы обеспечить отжиг комплементарных повторяющихся последовательностей. После отжига одноцепочечные фрагменты выступающих концов отщепляются. Новый синтез ДНК заполняет любые пробелы, а лигирование восстанавливает дуплекс ДНК. В результате обработки последовательность ДНК между двумя повторами удаляется. Длина делеции может зависеть от многих факторов, включая расположение двух использованных повторов и путь или процессивность разреза.
[0791] В отличие от путей HDR, SSA не требуется матричная нуклеиновая кислота для изменения или коррекции последовательности нуклеиновой кислоты-мишени. Вместо этого используется комплементарная повторяющаяся последовательность.
(5) Другие пути репарации ДНК
(a) SSBR (репарация одноцепочечного разрыва)
[0792] Одноцепочечные разрывы (SSB) в геноме подвергаются репарации путем SSBR, который является механизмом, отличным от механизмов репарации DSB, которые обсуждались выше. Путь SSBR имеет четыре основные стадии: детектирование SSB, обработка концов ДНК, заполнение пробела ДНК и лигирование ДНК. Более подробное объяснение дано в Caldecott, Nature Reviews Genetics 9, 619-631 (август 2008 г.), а краткое изложение приведено в настоящем описании.
[0793] На первой стадии, когда формируется SSB, PARP1 и/или PARP2 распознают разрыв и рекрутируют аппарат репарации. Связывание и активность PARP1 при разрывах ДНК являются временными, и, по-видимому, они ускоряют SSBr, стимулируя очаговое накопление или стабильность белковых комплексов SSBr в очаге поражения. Возможно, наиболее важным из этих белков SSBr является XRCC1, который функционирует в качестве молекулярного каркаса, который взаимодействует, стабилизирует и стимулирует множество ферментативных компонентов процесса SSBr, включая белок, ответственный за очистку 3'- и 5'-концов ДНК. Например, XRCC1 взаимодействует с несколькими белками (ДНК-полимераза бета, PNK и тремя нуклеазами, APE1, APTX и APLF), которые способствуют процессированию концов. APE1 обладает эндонуклеазной активностью. APLF проявляет эндонуклеазную и 3'-5'-экзонуклеазную активность. APTX обладает эндонуклеазной и 3'-5'-экзонуклеазной активностью.
[0794] Это концевое процессирование является важной стадией SSBR, поскольку 3'- и/или 5'-концы большинства, если не всех, SSB «повреждены». Процессирование концов обычно включает восстановление поврежденного 3'-конца до гидроксилированного состояния и/или поврежденного 5'-конца до фосфатного фрагмента, так что концы становятся пригодными для лигирования. Ферменты, которые могут процессировать поврежденные 3'-концы, включают PNKP, APE1 и TDP1. Ферменты, которые могут процессировать поврежденные 5'-концы, включают PNKP, бета-ДНК-полимеразу и APTX. LIG3 (ДНК-лигаза III) также может участвовать в процессировании концов. Как только концы очищены, может произойти заполнение пробела.
[0795] На стадии заполнения пробела ДНК обычно присутствуют белки PARP1, ДНК-полимераза бета, XRCC1, FEN1 (флэп-эндонуклеаза 1), ДНК-полимераза дельта/эпсилон, PCNA и LIG1. Существует два способа заполнения пробелов: репарация короткими и длинными последовательностями. Репарация короткими последовательностями включает вставку единственного нуклеотида, который отсутствует. В некоторых SSB «заполнение пробелов» может продолжать вытеснять два или более нуклеотидов (сообщалось о смещении до 12 оснований). FEN1 является эндонуклеазой, которая удаляет смещенные 5'-остатки. Множество ДНК-полимераз, включая Pol β, участвуют в репарации SSB, причем выбор ДНК-полимеразы зависит от источника и типа SSB.
[0796] На четвертой стадии ДНК-лигаза, такая как LIG1 (Лигаза I) или LIG3 (Лигаза III), катализирует соединение концов. Репарация короткими последовательностями использует Лигазу III, а репарация длинными последовательностями использует Лигазу I.
[0797] Иногда SSBR связан с репликацией. Этот путь может включать один или более из CtIP, MRN, ERCC1 и FEN1. Дополнительные факторы, которые могут стимулировать SSBR, включают: aPARP, PARP1, PARP2, PARG, XRCC1, ДНК-полимеразу b, ДНК-полимеразу d, ДНК-полимеразу e, PCNA, LIG1, PNK, PNKP, APE1, APTX, APLF, TDP1, LIG3, FEN1, CtIP, MRN и ERCC1.
(b) MMR (репарация ошибочного спаривания)
[0798] Клетки содержат три пути эксцизионной репарации: MMR, BER и NER. Пути эксцизионной репарации имеют общую особенность в том, что они обычно распознают поражение на одной цепи ДНК, затем экзо/эндонуклеазы удаляют поражение и оставляют 1-30-нуклеотидный прбел, который последовательно заполняются ДНК-полимеразой и, наконец, запечатывается лигазой. Более полная картина дана в Li, Cell Research (2008) 18: 85-98, а краткое изложение представлено в настоящем описании.
[0799] Репарация ошибочного спаривания (MMR) работает с ошибочными основаниями ДНК.
[0800] Оба комплекса MSH2/6 или MSH2/3 обладают активностью АТФаз, которая играет важную роль в распознавании ошибочного спаривания и инициации репарации. MSH2/6 предпочтительно распознает ошибочное спаривание оснований и идентифицирует ошибочные пары из 1 или 2 нуклеотидов, в то время как MSH2/3 преимущественно распознает ошибочные пары более крупных ID.
[0801] hMLH1 гетеродимеризуется с hPMS2 с образованием hMutLα, который обладает активностью АТФазы и важен для нескольких стадий MMR. Он обладает PCNA/фактор репликации C (RFC)-зависимой эндонуклеазной активностью, которая играет важную роль в 3'-ник-направленной MMR с участием EXO1. (EXO1 является участником как HR, так и MMR.) Он регулирует терминацию вызванного ошибочным спариванием удаления. Лигаза I является соответствующей лигазой для этого пути. Дополнительные факторы, которые могут способствовать MMR, включают: EXO1, MSH2, MSH3, MSH6, MLH1, PMS2, MLH3, ДНК Pol d, RPA, HMGB1, RFC и ДНК-лигазу I.
(c) Эксцизионная репарация оснований (BER)
[0802] Путь эксцизионной репарации оснований (BER) активен на протяжении всего клеточного цикла; он отвечает прежде всего за удаление небольших, не искажающих спираль повреждений оснований из генома. Напротив, связанный с этим путь эксцизионной репарации нуклеотидов (обсуждаемый в следующем разделе) восстанавливает объемные повреждения, повреждающие спираль. Более подробное объяснение дано в Caldecott, Nature Reviews Genetics 9, 619-631 (август 2008 г.), а краткое изложение приведено в настоящем описании.
[0803] При повреждении основания ДНК инициируется эксцизионная репарация оснований (BER), и процесс может быть упрощен до пяти основных стадий: (a) удаление поврежденного основания ДНК; (b) разрез последующего основного сайта; (c) зачистка концов ДНК; (d) вставка корректного нуклеотида в восстанавливаемый пробел; и (e) лигирование оставшегося одноцепочечного разрыва в остове ДНК. Эти последние стадии похожи на SSBR.
[0804] На первой стадии ДНК-гликозилаза, специфичная для повреждения, иссекает поврежденное основание посредством расщепления N-гликозидной связи, связывающей основание с сахарофосфатным остовом. Затем AP-эндонуклеаза-1 (APE1) или бифункциональные ДНК-гликозилазы с ассоциированной лиазной активностью разрезают фосфодиэфирный остов для создания одноцепочечного разрыва ДНК (SSB). Третья стадия BER включает зачистку концов ДНК. Четвертая стадия в BER проводится с помощью Pol β, которая добавляет новый комплементарный нуклеотид в восстанавливаемый пробел, а на последней стадии XRCC1/Лигаза III запечатывает оставшийся одноцепочечный разрыв в остове ДНК. Это завершает путь репарации короткими последовательностями BER, в котором восстанавливается большинство (~ 80%) поврежденных оснований ДНК. Однако, если 5'-концы на стадии 3 устойчивы к активности процессинга концов, после одной вставки нуклеотида с помощью Pol β происходит переключение полимеразы на репликативные ДНК-полимеразы, Pol δ/ɛ, которые затем добавляют еще ~ 2-8 нуклеотидов в пробел репарации ДНК. Это создает 5'-флэп структуру, которая распознается и иссекается флэп-эндонуклеазой-1 (FEN-1) в ассоциации с фактора процессивности в виде ядерного антигена клеточной пролиферации (PCNA). Затем ДНК-лигаза I запечатывает оставшийся одноцепочечный разрыв в остове ДНК и завершает BER длинными последовательностями. Дополнительные факторы, которые могут способствовать пути BER, включают: ДНК-гликозилазу, APE1, Polb, Pold, Pole, XRCC1, Лигазу III, FEN-1, PCNA, RECQL4, WRN, MYH, PNKP и APTX.
(d) Эксцизионная репарация нуклеотидов (NER)
[0805] Эксцизионная репарация нуклеотидов (NER) является важным эксцизионным механизмом, который удаляет объемные повреждения, нарушающие спираль, из ДНК. Дополнительные подробности о NER приведены в Marteijn et al., Nature Reviews, Molecular Cell Biology 15, 465-481 (2014), а краткое изложение приведено в настоящем описании. NER - расширенный путь, охватывающий два меньших пути: глобальный геномный NER (GG-NER) и связанная с транскрипцией репарация NER (TC-NER). GG-NER и TC-NER используют разные факторы для распознавания повреждений ДНК. Однако, они используют один и тот же аппарат для разреза поражения, репарации и лигирования.
[0806] Как только повреждение распознано, клетка удаляет короткий одноцепочечный сегмент ДНК, содержащий повреждение. Эндонуклеазы XPF/ERCC1 и XPG (кодируемые ERCC5) удаляют поражение, разрезая поврежденную цепь с обеих сторон поражения, что приводит к одноцепочечному пробелу из 22-30 нуклеотидов. Затем клетка выполняет синтез, заполняющий пробел ДНК и лигирование. В этом процессе участвуют: PCNA, RFC, ДНК Пол. δ, ДНК Пол. ε или ДНК Пол. κ и ДНК-лигаза I или XRCC1/Лигаза III. Реплицирующиеся клетки имеют тенденцию использовать ДНК пол. ε и ДНК-лигазу I, в то время как нереплицирующиеся клетки имеют тенденцию использовать ДНК пол. δ, ДНК пол. κ и комплекс XRCC1/Лигазы III для выполнения стадии лигирования.
[0807] NER может включать следующие факторы: XPA-G, POLH, XPF, ERCC1, XPA-G и LIG1. NER, связанная с транскрипцией, (TC-NER), может включать следующие факторы: CSA, CSB, XPB, XPD, XPG, ERCC1 и TTDA. Дополнительные факторы, которые могут способствовать пути репарации NER, включают XPA-G, POLH, XPF, ERCC1, XPA-G, LIG1, CSA, CSB, XPA, XPB, XPC, XPD, XPF, XPG, TTDA, UVSSA, USP7, CETN2, RAD23B, UV-DDB, подкомплекс CAK, RPA и PCNA.
(e) Внутрицепочечная поперечная сшивка (ICL)
[0808] Специальный путь, называемый ICL, восстанавливает межцепочечные поперечные сшивки. Межцепочечные поперечные сшивки или ковалентные поперечные сшивки между основаниями в разных цепях ДНК могут происходить во время репликации или транскрипции. Репарация ICL включает в себя координацию множества процессов репарации, в частности, нуклеолитическую активность, синтеза «чрез/сквозь» повреждения (TLS) и HDR. Нуклеазы рекрутируют для удаления ICL с обеих сторон поперечно сшитых оснований, в то время как TLS и HDR координируют для репарации разрезанных цепей. Репарация ICL может включать следующие факторы: эндонуклеазы, например, XPF и RAD51C, эндонуклеазы, такие как RAD51, полимеразы "чрез/сквозь" повреждение, например, ДНК-полимераза дзета и Rev1), и белки анемии Фанкони (FA), например, FancJ.
(f) Другие пути
[0809] У млекопитающих существует несколько других путей репарации ДНК.
[0810] Синтез «чрез/сквозь» повреждение (TLS) представляет собой путь для репарации одноцепочечного разрыва, оставшегося после дефектного события репликации, и включает в себя полимеразы "чрез/сквозь" повреждение, например, ДНК пол. ζ и Rev1.
[0811] Безошибочная пострепликативная репарация (PRR) - это еще один путь репарации одноцепочечного разрыва, оставшегося после дефектного события репликации.
(6) Примеры гРНК в методах редактирования генома
[0812] Любую из молекул гРНК, как описано в настоящем документе, можно использовать с любыми молекулами Cas9, которые генерируют двухцепочечный разрыв или одноцепочечный разрыв, для изменения последовательности нуклеиновой кислоты-мишени, например положения-мишени или генетической сигнатуры-мишени. В некоторых примерах нуклеиновая кислота-мишень находится в локусах TRAC, TRBC1 и/или TRBC2 или рядом с ними, таких как любые из описанных. В некоторых вариантах осуществления молекула рибонуклеиновой кислоты, такая как молекула гРНК, и белок, такой как белок Cas9 или его варианты, вводятся в любую из сконструированных клеток, предложенных в настоящем описании. Молекулы гРНК, используемые в этих способах, описаны ниже.
[0813] В одном из вариантов осуществления, гРНК, например химерная гРНК, имеет такую конфигурацию, что она имеет одно или более из следующих свойств;
а) она может позиционироваться, например, при нацеливании молекулы Cas9, которая делает двухцепочечные разрывы, двухцепочечный разрыв (i) в пределах 50, 100, 150, 200, 250, 300, 350, 400, 450 или 500 нуклеотидов положения-мишени или (ii) достаточно близко, чтобы положение-мишень находилось в области резекции концов;
b) она имеет нацеливающий домен по меньшей мере из 16 нуклеотидов, например, нацеливающий домен из (i) 16, (ii), 17, (iii) 18, (iv) 19, (v) 20, (vi) 21, (vii) 22, (viii) 23, (ix) 24, (x) 25 или (xi) 26 нуклеотидов; и
с)
(i) проксимальный и хвостовой домены, взятые вместе, содержат, по меньшей мере, 15, 18, 20, 25, 30, 31, 35, 40, 45, 49, 50 или 53 нуклеотида, например, по меньшей мере, 15, 18, 20, 25, 30, 31, 35, 40, 45, 49, 50 или 53 нуклеотида из встречающегося в природе S. pyogenes, S. thermophilus, S. aureus или N. meningitidis хвостового и проксимального домена или последовательности, которая отличается не более чем на 1, 2, 3, 4, 5; 6, 7, 8, 9 или 10 нуклеотидов из них;
(ii) имеется по меньшей мере 15, 18, 20, 25, 30, 31, 35, 40, 45, 49, 50 или 53 нуклеотида в 3'-области оносительно последнего нуклеотида второго домена комплементарности, например, по меньшей мере 15, 18, 20, 25, 30, 31, 35, 40, 45, 49, 50 или 53 нуклеотида из соответствующей последовательности встречающейся в природе гРНК S. pyogenes, S. thermophilus, S. aureus или N. meningitidis, или последовательности, которая отличается от нее не более чем на 1, 2, 3, 4, 5; 6, 7, 8, 9 или 10 нуклеотидов;
(iii) имеется по меньшей мере 16, 19, 21, 26, 31, 32, 36, 41, 46, 50, 51 или 54 нуклеотида в 3'-области оносительно последнего нуклеотида второго домена комплементарности, например, по меньшей мере 16, 19, 21, 26, 31, 32, 36, 41, 46, 50, 51 или 54 нуклеотида из соответствующей последовательности встречающейся в природе гРНК S. pyogenes, S. thermophilus, S. aureus или N. meningitidis или последовательности, которая отличается от нее не более чем на 1, 2, 3, 4, 5; 6, 7, 8, 9 или 10 нуклеотидов;
(iv) хвостовой домен имеет длину, по меньшей мере, 10, 15, 20, 25, 30, 35 или 40 нуклеотидов, например, он содержит, по меньшей мере, 10, 15, 20, 25, 30, 35 или 40 нуклеотидов из встречающегося в природе хвостового домена S. pyogenes, S. thermophilus, S. aureus или N. meningitidis, или последовательность, которая отличается от него не более чем на 1, 2, 3, 4, 5; 6, 7, 8, 9 или 10 нуклеотидов; или
(v) хвостовой домен содержит 15, 20, 25, 30, 35, 40 нуклеотидов или все из соответствующих частей встречающегося в природе хвостового домена, например встречающегося в природе хвостового домена S. pyogenes, S. thermophilus, S. aureus или Н. meningitidis.
[0814] В одном варианте осуществления гРНК сконфигурирована так, что она включает свойства: a и b (i). В одном варианте осуществления гРНК сконфигурирована так, что она включает свойства: a и b (ii). В одном варианте осуществления гРНК сконфигурирована так, что она включает свойства: a и b (iii). В одном варианте осуществления гРНК сконфигурирована так, что она включает свойства: a и b (iv). В одном варианте осуществления гРНК сконфигурирована так, что она включает свойства: a и b (v). В одном варианте осуществления гРНК сконфигурирована так, что она включает свойства: a и b (vi). В одном варианте осуществления гРНК сконфигурирована так, что она включает свойства: a и b (vii). В одном варианте осуществления гРНК сконфигурирована так, что она включает свойства: a и b (viii). В одном варианте осуществления гРНК сконфигурирована так, что она включает свойства: a и b (ix). В одном варианте осуществления гРНК сконфигурирована так, что она включает свойства: a и b (x). В одном варианте осуществления гРНК сконфигурирована так, что она включает свойства: a и b (xi). В одном варианте осуществления гРНК сконфигурирована так, что она включает свойства: a и c. В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a, b и c. В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (i) и c (i). В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (i) и c (ii). В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (ii) и c (i). В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (ii) и c (ii). В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (iii) и c (i). В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (iii) и c (ii). В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (iv) и c (i). В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (iv) и c (ii). В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (v) и c (i). В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (v) и c (ii). В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (vi) и c (i). В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (vi) и c (ii). В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (vii) и c (i). В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (vii) и c (ii). В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (viii) и c (i). В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (viii) и c (ii). В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (ix) и c (i). В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (ix) и c (ii). В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (x) и c (i). В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (x) и c (ii). В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (xi) и c (i). В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (xi) и c (ii).
[0815] В одном варианте осуществления, гРНК, например химерная гРНК, имеет такую конфигурацию, что она имеет одно или более из следующих свойств;
а) одна или обе из гРНК могут позиционироваться, например, при нацеливании молекулы Cas9, которая делает одноцепочечные разрывы, одноцепочечный разрыв в пределах (i) 50, 100, 150, 200, 250, 300, 350, 400, 450, или 500 нуклеотидов от положения-мишени или (ii) достаточно близко, чтобы положение-мишень находилось в области резекции концов;
b) одна или обе имеют нацеливающий домен по меньшей мере из 16 нуклеотидов, например, нацеливающий домен из (i) 16, (ii), 17, (iii) 18, (iv) 19, (v) 20, (vi) 21, (vii) 22, (viii) 23, (ix) 24, (x) 25 или (xi) 26 нуклеотидов; и
с)
(i) проксимальный и хвостовой домены, взятые вместе, содержат, по меньшей мере, 15, 18, 20, 25, 30, 31, 35, 40, 45, 49, 50 или 53 нуклеотида, например, по меньшей мере, 15, 18, 20, 25, 30, 31, 35, 40, 45, 49, 50 или 53 нуклеотида из встречающегося в природе S. pyogenes, S. thermophilus, S. aureus или N. meningitidis хвостового и проксимального домена или последовательности, которая отличается не более чем на 1, 2, 3, 4, 5; 6, 7, 8, 9 или 10 нуклеотидов из них;
(ii) имеется по меньшей мере 15, 18, 20, 25, 30, 31, 35, 40, 45, 49, 50 или 53 нуклеотида в 3'-области оносительно последнего нуклеотида второго домена комплементарности, например, по меньшей мере 15, 18, 20, 25, 30, 31, 35, 40, 45, 49, 50 или 53 нуклеотида из соответствующей последовательности встречающейся в природе гРНК S. pyogenes, S. thermophilus, S. aureus или N. meningitidis, или последовательности, которая отличается от нее не более чем на 1, 2, 3, 4, 5; 6, 7, 8, 9 или 10 нуклеотидов;
(iii) имеется по меньшей мере 16, 19, 21, 26, 31, 32, 36, 41, 46, 50, 51 или 54 нуклеотида в 3'-области оносительно последнего нуклеотида второго домена комплементарности, например, по меньшей мере 16, 19, 21, 26, 31, 32, 36, 41, 46, 50, 51 или 54 нуклеотида из соответствующей последовательности встречающейся в природе гРНК S. pyogenes, S. thermophilus, S. aureus или N. meningitidis или последовательности, которая отличается от нее не более чем на 1, 2, 3, 4, 5; 6, 7, 8, 9 или 10 нуклеотидов;
(iv) хвостовой домен имеет длину, по меньшей мере, 10, 15, 20, 25, 30, 35 или 40 нуклеотидов, например, он содержит, по меньшей мере, 10, 15, 20, 25, 30, 35 или 40 нуклеотидов из встречающегося в природе хвостового домена S. pyogenes, S. thermophilus, S. aureus или N. meningitidis, или последовательность, которая отличается от него не более чем на 1, 2, 3, 4, 5; 6, 7, 8, 9 или 10 нуклеотидов; или
(v) хвостовой домен содержит 15, 20, 25, 30, 35, 40 нуклеотидов или все из соответствующих частей встречающегося в природе хвостового домена, например встречающегося в природе хвостового домена S. pyogenes, S. thermophilus, S. aureus или Н. meningitidis.
[0816] В одном варианте осуществления гРНК сконфигурирована так, что она включает свойства: a и b (i). В одном варианте осуществления гРНК сконфигурирована так, что она включает свойства: a и b (ii). В одном варианте осуществления гРНК сконфигурирована так, что она включает свойства: a и b (iii). В одном варианте осуществления гРНК сконфигурирована так, что она включает свойства: a и b (iv). В одном варианте осуществления гРНК сконфигурирована так, что она включает свойства: a и b (v). В одном варианте осуществления гРНК сконфигурирована так, что она включает свойства: a и b (vi). В одном варианте осуществления гРНК сконфигурирована так, что она включает свойства: a и b (vii). В одном варианте осуществления гРНК сконфигурирована так, что она включает свойства: a и b (viii). В одном варианте осуществления гРНК сконфигурирована так, что она включает свойства: a и b (ix). В одном варианте осуществления гРНК сконфигурирована так, что она включает свойства: a и b (x). В одном варианте осуществления гРНК сконфигурирована так, что она включает свойства: a и b (xi). В одном варианте осуществления гРНК сконфигурирована так, что она включает свойства: a и c. В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a, b и c. В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (i) и c (i). В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (i) и c (ii). В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (ii) и c (i). В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (ii) и c (ii). В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (iii) и c (i). В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (iii) и c (ii). В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (iv) и c (i). В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (iv) и c (ii). В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (v) и c (i). В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (v) и c (ii). В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (vi) и c (i). В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (vi) и c (ii). В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (vii) и c (i). В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (vii) и c (ii). В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (viii) и c (i). В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (viii) и c (ii). В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (ix) и c (i). В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (ix) и c (ii). В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (x) и c (i). В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (x) и c (ii). В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (xi) и c (i). В одном варианте осуществления гРНК сконфигурирована так, что включает свойства: a (i), b (xi) и c (ii).
[0817] В одном варианте осуществления гРНК используется с молекулой никазы Cas9, обладающей активностью HNH, например молекулой Cas9, обладающей инактивированной активностью RuvC, например, с молекулой Cas9, имеющей мутацию в D10, например мутацию D10A.
[0818] В одном варианте осуществления гРНК используется с молекулой никазы Cas9, обладающей активностью RuvC, например молекулой Cas9, обладающей инактивированной активностью HNH, например, с молекулой Cas9, имеющей мутацию в H840, например H840A.
[0819] В одном варианте осуществления пара гРНК, например пара химерных гРНК, содержащих первую и вторую гРНК, сконфигурирована так, что они имеют одно или более из следующих свойств;
а) одна или обе из гРНК могут позиционироваться, например, при нацеливании молекулы Cas9, которая делает одноцепочечные разрывы, одноцепочечный разрыв в пределах (i) 50, 100, 150, 200, 250, 300, 350, 400, 450, или 500 нуклеотидов от положения-мишени или (ii) достаточно близко, чтобы положение-мишень находилось в области резекции концов;
b) одна или обе имеют нацеливающий домен по меньшей мере из 16 нуклеотидов, например, нацеливающий домен из (i) 16, (ii), 17, (iii) 18, (iv) 19, (v) 20, (vi) 21, (vii) 22, (viii) 23, (ix) 24, (x) 25 или (xi) 26 нуклеотидов;
c) для одной или обеих:
(i) проксимальный и хвостовой домены, взятые вместе, содержат, по меньшей мере, 15, 18, 20, 25, 30, 31, 35, 40, 45, 49, 50 или 53 нуклеотида, например, по меньшей мере, 15, 18, 20, 25, 30, 31, 35, 40, 45, 49, 50 или 53 нуклеотида из встречающегося в природе S. pyogenes, S. thermophilus, S. aureus или N. meningitidis хвостового и проксимального домена или последовательности, которая отличается не более чем на 1, 2, 3, 4, 5; 6, 7, 8, 9 или 10 нуклеотидов из них;
(ii) имеется по меньшей мере 15, 18, 20, 25, 30, 31, 35, 40, 45, 49, 50 или 53 нуклеотида в 3'-области оносительно последнего нуклеотида второго домена комплементарности, например, по меньшей мере 15, 18, 20, 25, 30, 31, 35, 40, 45, 49, 50 или 53 нуклеотида из соответствующей последовательности встречающейся в природе гРНК S. pyogenes, S. thermophilus, S. aureus или N. meningitidis, или последовательности, которая отличается от нее не более чем на 1, 2, 3, 4, 5; 6, 7, 8, 9 или 10 нуклеотидов;
(iii) имеется по меньшей мере 16, 19, 21, 26, 31, 32, 36, 41, 46, 50, 51 или 54 нуклеотида в 3'-области оносительно последнего нуклеотида второго домена комплементарности, например, по меньшей мере 16, 19, 21, 26, 31, 32, 36, 41, 46, 50, 51 или 54 нуклеотида из соответствующей последовательности встречающейся в природе гРНК S. pyogenes, S. thermophilus, S. aureus или N. meningitidis или последовательности, которая отличается от нее не более чем на 1, 2, 3, 4, 5; 6, 7, 8, 9 или 10 нуклеотидов;
(iv) хвостовой домен имеет длину, по меньшей мере, 10, 15, 20, 25, 30, 35 или 40 нуклеотидов, например, он содержит, по меньшей мере, 10, 15, 20, 25, 30, 35 или 40 нуклеотидов из встречающегося в природе хвостового домена S. pyogenes, S. thermophilus, S. aureus или N. meningitidis; или последовательность, которая отличается от них не более чем на 1, 2, 3, 4, 5; 6, 7, 8, 9 или 10 нуклеотидов; или
(v) хвостовой домен содержит 15, 20, 25, 30, 35, 40 нуклеотидов или все из соответствующих частей встречающегося в природе хвостового домена, например встречающегося в природе хвостового домена S. pyogenes, S. thermophilus, S. aureus, или N. meningitidis.
d) гРНК имеют такую конфигурацию, что при гибридизации с нуклеиновой кислотой-мишенью они разделяются на 0-50, 0-100, 0-200, по меньшей мере, 10, по меньшей мере, 20, по меньшей мере, 30 или, по меньшей мере, 50 нуклеотидов;
e) разрывы, сделанные первой и второй гРНК, находятся на разных цепях; и
f) PAM обращены наружу.
[0820] В варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a и b (i). В одном варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a и b (ii). В одном варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a и b (iii). В одном варианте осуществления одна или обе гРНК сконфигурированы так, что они включают свойства: a и b (iv). В одном варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a и b (v). В одном варианте осуществления одна или обе гРНК сконфигурированы так, что они включают свойства: a и b (vi). В одном варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a и b (vii). В одном варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a и b (viii). В варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a и b (ix). В одном варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a и b (x). В одном варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a и b (xi). В одном варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a и c. В варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a, b и c. В варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (i) и c (i). В варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (i) и c (ii). В варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (i), c и d. В варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (i), c и e. В варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (i), c, d и e. В одном варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (ii) и c (i). В одном варианте осуществления одна или обе из этих гРНК сконфигурированы так, что они включают свойства: a (i), b (ii) и c (ii). В варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (ii), c и d. В варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (ii), c и e. В варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (ii), c, d и e. В одном варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (iii) и c (i). В одном варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (iii) и c (ii). В варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (iii), c и d. В варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (iii), c и e. В варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (iii), c, d и e. В варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (iv) и c (i). В варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (iv) и c (ii). В варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (iv), c и d. В одном варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (iv), c и e. В одном варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (iv), c, d и e. В одном варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (v) и c (i). В одном варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (v) и c (ii). В одном варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (v), c и d. В варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (v), c и e. В варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (v), c, d и e. В одном варианте осуществления одна или обе гРНК сконфигурированы так, что они включают свойства: a (i), b (vi) и c (i). В одном варианте осуществления одна или обе из этих гРНК сконфигурированы так, что они включают свойства: a (i), b (vi) и c (ii). В варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (vi), c и d. В варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (vi), c и e. В варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (vi), c, d и e. В варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (vii) и c (i). В одном варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (vii) и c (ii). В варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (vii), c и d. В одном варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (vii), c и e. В варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (vii), c, d и e. В одном варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (viii) и c (i). В одном варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (viii) и c (ii). В одном варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (viii), c и d. В одном варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (viii), c и e. В одном варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (viii), c, d и e. В варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (ix) и c (i). В одном варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (ix) и c (ii). В варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (ix), c и d. В варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (ix), c и e. В одном варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (ix), c, d и e. В варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (x) и c (i). В одном варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (x) и c (ii). В варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (x), c и d. В варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (x), c и e. В варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (x), c, d и e. В варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (xi) и c (i). В одном варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (xi) и c (ii). В варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (xi), c и d. В варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (xi), c и e. В варианте осуществления одна или обе из гРНК сконфигурированы так, что они включают свойства: a (i), b (xi), c, d и e.
[0821] В одном варианте осуществления гРНК используются с молекулой никазы Cas9, обладающей активностью HNH, например молекулой Cas9, обладающей активностью RuvC, например молекулой Cas9, имеющей мутацию в D10, например мутацию D10A.
[0822] В одном варианте осуществления гРНК используются с молекулой никазы Cas9, обладающей активностью RuvC, например молекулой Cas9, обладающей инактивированной активностью HNH, например молекулой Cas9, имеющей мутацию в H840, например H840A. В одном варианте осуществления гРНК используются с молекулой никазы Cas9, обладающей активностью RuvC, например молекулой Cas9, обладающей инактивированной активностью HNH, например молекулой Cas9, имеющей мутацию в N863, например, N863A.
(7) Функциональный анализ агентов для редактирования генов
[0823] Любая из молекул Cas9, молекулы гРНК, комплексы молекулы Cas9/молекулы гРНК могут быть оценены известными в данной области способами или как описано в настоящем документе. Например, иллюстративные способы оценки эндонуклеазной активности молекулы Cas9 описаны, например, в Jinek et al., Science 2012, 337 (6096): 816-821.
(a) Анализ связывания и расщепления: тестирование эндонуклеазной активности молекулы Cas9
[0824] Способность комплекса молекула Cas9/молекула гРНК связываться и расщеплять нуклеиновую кислоту-мишень может оцениваться в анализе расщепления плазмиды. В этом анализе синтетическую или in vitro-транскрибированную молекулу гРНК предварительно отжигают перед реакцией, нагревая до 95°C и медленно охлаждая до комнатной температуры. Нативную или линеаризованную рестрикционным гидролизом плазмидную ДНК (300 нг (~ 8 нМ)) инкубируют в течение 60 мин при 37°С с очищенной молекулой белка Cas9 (50-500 нМ) и гРНК (50-500 нМ, 1:1) в буфере расщепления плазмиды Cas9 (20 мМ HEPES, рН 7,5, 150 мМ KCl, 0,5 мМ DTT, 0,1 мМ EDTA) с или без 10 мМ MgCl2. Реакции останавливают 5-кратным ДНК-загрузочным буфером (30% глицерина, 1,2% SDS, 250 мМ EDTA), разделяют на электрофорезе в 0,8 или 1% -ном агарозном геле и визуализируют окрашиванием этидийбромидом. Полученные продукты расщепления выявляют, расщепляет ли молекула Cas9 обе цепи ДНК или только одну из двух цепей. Например, линейные продукты ДНК указывают на расщепление обеих цепей ДНК. Разорванные незамкнутые кольцевые продукты выявляют, что расщепляется только одна из двух цепей.
[0825] Альтернативно, способность комплекса молекулы Cas9/молекулы гРНК связываться и расщеплять нуклеиновую кислоту-мишень может быть оценена в анализе расщепления олигонуклеотидной ДНК. В этом анализе олигонуклеотиды ДНК (10 пмоль) радиоактивно метят путем инкубации с 5 единицами полинуклеотидкиназы T4 и ~ 3-6 пмоль (~ 20-40 мКи) [γ-32P] -АТФ в 1X реакционном буфере T4 полинуклеотидкиназы при 37°С в течение 30 мин, в объеме реакции 50 мкл. После тепловой инактивации (65°С в течение 20 мин) реакционные смеси очищают через колонку для удаления неинкорпорированной метки. Дуплексные субстраты (100 нМ) получают путем отжига меченых олигонуклеотидов с эквимолярным количеством немеченого комплементарного олигонуклеотида при 95°С в течение 3 минут с последующим медленным охлаждением до комнатной температуры. Для анализа расщепления молекулы гРНК отжигают путем нагревания до 95°С в течение 30 с с последующим медленным охлаждением до комнатной температуры. Cas9 (конечная концентрация 500 нМ) предварительно инкубируют с отожженными молекулами гРНК (500 нМ) в буфере для анализа расщепления (20 мМ HEPES, рН 7,5, 100 мМ KCl, 5 мМ MgCl2, 1 мМ DTT, 5% глицерина) в общем количестве объем 9 мкл. Реакции инициируют добавлением 1 мкл ДНК-мишени (10 нМ) и инкубируют в течение 1 ч при 37°С. Реакции гасят добавлением 20 мкл загрузочного красителя (5 мМ ЭДТА, 0,025% SDS, 5% глицерина в формамиде) и нагревают до 95°С в течение 5 минут. Продукты расщепления разделяют на 12% денатурирующих полиакриламидных гелях, содержащих 7 М мочевины, и визуализируют путем формирования изображения на люминисцентном фосфорном покрытии. Полученные в результате продукты расщепления выявляют, расщеплена ли комплементарная цепь, некомплементарная цепь или они обе.
[0826] Один или оба из этих анализов могут быть использованы для оценки пригодности любой предложенной молекулы гРНК или молекулы Cas9.
(b) Анализ связывания: тестирование связывания молекулы Cas9 с ДНК-мишенью
[0827] Иллюстративные способы оценки связывания молекулы Cas9 с ДНК-мишенью описаны, например, в Jinek et al., Science 2012; 337 (6096): 816-821.
[0828] Например, в анализе изменения электрофоретической подвижности дуплексы ДНК-мишени образуются путем смешивания каждой цепи (10 нмоль) в деионизированной воде, нагревания до 95°С в течение 3 минут и медленного охлаждения до комнатной температуры. Все ДНК очищают на 8% нативных гелях, содержащих 1X TBE. Полосы ДНК визуализируют с помощью ультрафиолетового затенения, иссекают и элюируют путем вымачивания кусочков геля в обработанной DEPC H2O. Элюированную ДНК осаждают этанолом и растворяют в обработанной DEPC H2O. Образцы ДНК с 5'-конца мечены [γ-32P] -АТФ с использованием Т4-полинуклеотидкиназы в течение 30 мин при 37°С. Полинуклеотидкиназу денатурируют нагреванием при 65°С в течение 20 мин, и неинкорпорированную радиоактивную метку удаляют с использованием колонки. Анализы связывания проводят в буфере, содержащем 20 мМ HEPES, рН 7,5, 100 мМ KCl, 5 мМ MgCl2, 1 мМ DTT и 10% глицерина в общем объеме 10 мкл. Молекула белка Cas9 программируется с помощью эквимолярных количеств предварительно отожженной молекулы гРНК и титруется от 100 пМ до 1 мкМ. ДНК с радиоактивной меткой добавляют до конечной концентрации 20 пМ. Образцы инкубируют в течение 1 часа при 37°С и растворяют при 4°С в 8% нативном полиакриламидном геле, содержащем 1X TBE и 5 мМ MgCl2. Гели сушат и ДНК визуализируют путем формирования изображения на люминисцентном фосфорном покрытии.
(c) Методы измерения термостабильности комплексов Cas9/гРНК
[0829] Термостабильность комплексов рибонуклеопротеина (RNP) Cas9-гРНК можно определить с помощью дифференциальной сканирующей флуориметрии (DSF) и других методов. Термостабильность белка может повышаться при благоприятных условиях, таких как добавление связывающей молекулы РНК, например, гРНК. Таким образом, информация, касающаяся термостабильности комплекса Cas9/gRNA, полезна для определения стабильности комплекса.
(d) Дифференциальная сканирующая флуориметрия (DSF)
[0830] Термостабильность комплексов рибонуклеопротеина (RNP) Cas9-гРНК может быть измерена с помощью DSF. Комплексы RNP, как описано ниже, включают последовательность рибонуклеотидов, таких как РНК или гРНК, и белок, такой как белок Cas9 или его вариант. Этот метод измеряет термостабильность белка, которая может увеличиваться при благоприятных условиях, таких как добавление молекулы связывающей РНК, например, гРНК.
[0831] Анализ может быть применен несколькими способами. Иллюстративные протоколы включают, но не ограничиваются ими, протокол для определения целевых условий раствора для образования RNP (анализ 1, см. ниже), протокол для тестирования целевого стехиометрического соотношения гРНК: белок Cas9 (анализ 2, см. ниже), протокол для скрининга эффективных молекул гРНК для молекул Cas9, например молекул дикого типа или мутантных молекул Cas9 (анализ 3, см. ниже), и протокол для изучения образования RNP в присутствии ДНК-мишени (анализ 4). В некоторых вариантах осуществления анализ проводится с использованием двух разных протоколов, один для проверки наилучшего стехиометрического соотношения гРНК: белок Cas9, а другой для определения наилучших условий раствора для образования RNP.
[0832] Чтобы определить наилучший раствор для формирования комплексов RNP, готовят раствор 2 мкМ Cas9 в воде+10x SYPRO Orange® (Life Technologies cat # S-6650) и распределяют в 384-луночный планшет. Затем добавляют эквимолярное количество гРНК, разведенное в растворах с различным рН и солью. После инкубации при комнатной температуре в течение 10 минут и кратковременного центрифугирования для удаления любых пузырьков используется термоциклер Bio-Rad CFX384™ Real-Time System C1000 Touch ™ с программным обеспечением Bio-Rad CFX Manager для запуска градиента от 20°C до 90°C с повышением температуры на 1°С каждые 10 секунд.
[0833] Второй анализ включает смешивание различных концентраций гРНК с 2 мкМ Cas9 в оптимальном буфере из анализа 1 выше и инкубации при комнатной температуре в течение 10 минут в 384-луночном планшете. Добавляют равный объем оптимального буфера+10x SYPRO Orange® (Life Technologies cat # S-6650) и планшет герметизируют клеем Microseal® B (MSB-1001). После непродолжительного центрифугирования для удаления пузырьков, термоциклер Bio-Rad CFX384™ Real-Time System C1000 Touch™ с программным обеспечением Bio-Rad CFX Manager используется для запуска градиента от 20°C до 90°C с увеличением на 1°С при температуре каждые 10 секунд.
[0834] В третьем анализе очищают представляющую интерес молекулу Cas9 (например, белок Cas9, например, вариантный белок Cas9). Библиотека вариантов молекул гРНК синтезируется и ресуспендируется до концентрации 20 мкМ. Молекула Cas9 инкубируется с молекулой гРНК в конечной концентрации 1 мкМ каждые в заранее определенном буфере в присутствии 5x SYPRO Orange® (Life Technologies cat # S-6650). После инкубации при комнатной температуре в течение 10 минут и центрифугирования при 2000 об/мин в течение 2 минут для удаления пузырьков для запуска градиента используется термоциклер Bio-Rad CFX384™ Real-Time System C1000 Touch ™ с программным обеспечением Bio-Rad CFX Manager для запуска градиента от 20°C до 90°C с повышением температуры на 1°C каждые 10 секунд.
[0835] В четвертом анализе DSF-эксперимент проводят со следующими образцами: только белок Cas9, белок Cas9 с гРНК, белок Cas9 с гРНК и ДНК-мишенью и белок Cas9 с ДНК-мишенью. Порядок смешивания компонентов: реакционный раствор, белок Cas9, гРНК, ДНК и SYPRO Orange. Реакционный раствор содержит 10 мМ HEPES, рН 7,5, 100 мМ NaCl, в отсутствие или в присутствии MgCl2. После центрифугирования при 2000 об/мин в течение 2 минут для удаления любых пузырьков используется термоциклер Bio-Rad CFX384™ Real-Time System C1000 Touch™ с программным обеспечением Bio-Rad CFX Manager для запуска градиента от 20°C до 90°C с повышением температуры на 1°С каждые 10 секунд.
b) Доставка агентов для генного редактирования
[0836] В некоторых вариантах осуществления уменьшение, делеция, устранение, нокаут или нарушение эндогенных генов, кодирующих TCR, таких как TRAC и TRBC1 или TRBC2, осуществляется путем доставки или введения одного или более агентов, способных к введению расщепления, например компонентов Cas9 и/или гРНК в клетку с использованием любого из ряда известных способов доставки или носителя для введения или переноса в клетки, например, с использованием лентивирусных векторов доставки, или любых известных способов или носителей для доставки молекул Cas9 и гРНК. Иллюстративные способы описаны, например, в Wang et al. (2012) J. Immunother. 35(9): 689-701; Cooper et al. (2003) Blood. 101:1637-1644; Verhoeyen et al. (2009) Methods Mol Biol. 506: 97-114; и Cavalieri et al. (2003) Blood. 102 (2): 497-505, WO 2015/161276; US 2015/0056705, US 2016/0272999, US 2017/0211075; или US 2017/0016027. В некоторых вариантах осуществления последовательности нуклеиновых кислот, кодирующие один или более компонентов одного или более агентов, способных вводить расщепление, например разрыв ДНК, вводят в клетки, например, любыми способами для введения нуклеиновых кислот в клетку, описанную в настоящем документе или известную В некоторых вариантах осуществления вектор, кодирующий компоненты одного или более агентов, способных вводить расщепление, такие как гид-РНК CRISPR и/или фермент Cas9, могут быть доставлены в клетку.
[0837] Молекулы Cas9 и молекулы гРНК, например комплекс молекула Cas9/молекула гРНК, могут быть использованы для манипуляций с клеткой, например, для редактирования нуклеиновой кислоты-мишени в широком спектре клеток.
[0838] В одном варианте осуществления клетка подвергается манипуляции путем редактирования (например, индуцирования мутации) одного или более генов-мишеней, например, как описано в настоящем документе. В некоторых вариантах осуществления модулируется экспрессия одного или более генов-мишеней (например, гена TRAC, TRBC1 и/или TRBC2). В другом варианте осуществления изобретения клетка подвергается манипуляции ex vivo путем редактирования (например, индукции мутации) одного или более генов-мишеней и/или модуляции экспрессии одного или более генов-мишеней, например гена TRAC, TRBC1 и/или TRBC2, и вводится пациенту. Источники клеток-мишеней для манипуляций ex vivo могут включать, например, кровь пациента, пуповинную кровь пациента или костный мозг пациента. Источники клеток-мишеней для манипуляций ex vivo могут также включать, например, гетерологичную донорскую кровь, пуповинную кровь или костный мозг.
[0839] Молекулы Cas9 и гРНК, описанные в настоящем документе, могут быть доставлены в клетку-мишень. В одном варианте осуществления клеткой-мишенью является Т-клетка, например, CD8+ Т-клетка (например, наивная CD8+ Т-клетка, Т-клетка центральной памяти или Т-клетка эффекторной памяти), CD4+ Т-клетка, Т-клетка-натуральный киллер (NKT-клетки), регуляторные T-клетки (Treg), T-клетки памяти стволовых клеток, лимфоидные клетки-предшественники, гемопоэтические стволовые клетки, клетки-натуральные киллеры (NK-клетки) или дендритные клетки. В одном варианте осуществления клетка-мишень представляет собой индуцированную плюрипотентную стволовую (iPS) клетку или клетку, полученную из iPS-клетки, например, iPS-клетку, сгенерированную у пациента, которую подвергают манипуляциям, чтобы изменить (например, вызвать мутацию) или манипулировать экспрессией одного или более генов-мишеней, например гена TRAC, TRBC1 и/или TRBC2, и она дифференцируется, например, в Т-клетку, например, в CD8+ Т-клетку (например, в наивную CD8+ Т-клетку, Т-клетку центральной памяти или эффекторную T-клетку памяти), CD4+ T-клетку, T-клетку памяти стволовых клеток, лимфоидную клетку-предшественник или гемопоэтическую стволовую клетку.
[0840] В одном варианте осуществления клетка-мишень была изменена так, чтобы она содержала гены специфических Т-клеточных рецепторов (TCR) (например, гены TRAC и TRBC). В другом варианте осуществления TCR обладает специфичностью связывания для опухолеассоциированного антигена, например, карциноэмбрионального антигена (CEA), GP100, антигена меланомы, распознаваемого Т-клетками 1 (MART1), антигена меланомы A3 (MAGEA3), NYESO1 или p53.
[0841] В одном варианте осуществления клетка-мишень была изменена, чтобы она содержала специфический химерный антигенный рецептор (CAR). В одном варианте осуществления CAR обладает специфичностью связывания для опухолеассоциированного антигена, например, CD19, CD20, карбоангидразы IX (CAIX), CD171, CEA, ERBB2, GD2, альфа-фолатного рецептора, антигена Льюиса Y, простат-специфического мембранного антигена (PSMA) или опухолеассоциированного гликопротеин 72 (TAG72).
[0842] В другом варианте осуществления клетка-мишень была изменена для связывания одного или более из следующих опухолевых антигенов, например, с помощью TCR или CAR. Опухолевые антигены могут включать, но не ограничиваются ими, AD034, AKT1, BRAP, CAGE, CDX2, CLP, CT-7, CT8/HOM-TES-85, cTAGE-1, Фибулин-1, HAGE, HCA587/MAGE-C2, hCAP-G, HCE661, HER2/neu, HLA-Cw, HOM-HD-21/Галектин9, HOM-MEEL-40/SSX2, HOM-RCC-3.1.3/CAXII, HOXA7, HOXB6, Hu, HUB1, KM -HN-3, KM-KN-1, KOC1, KOC2, KOC3, KOC3, LAGE-1, MAGE-1, MAGE-4a, MPP11, MSLN, NNP-1, NY-BR-1, NY-BR-62, NY-BR-85, NY-CO-37, NY-CO-38, NY-ESO-1, NY-ESO-5, NY-LU-12, NY-REN-10, NY-REN-19/LKB/STK11, NY-REN-21, NY-REN-26/BCR, NY-REN-3/NY-CO-38, NY-REN-33/SNC6, NY-REN-43, NY-REN-65, NY -REN-9, NY-SAR-35, OGFr, PLU-1, Rab38, RBPJkappa, RHAMM, SCP1, SCP-1, SSX3, SSX4, SSX5, TOP2A, TOP2B или тирозиназу.
(1) Методы Ex Vivo-доставки компонентов в клетки-мишени
[0843] Компоненты, например молекула Cas9 и молекула гРНК, могут быть введены в клетки-мишени в различных формах с использованием различных способов доставки и составов, см., например, Таблицы 19 и 20. Когда компонент Cas9 или гРНК кодируется в качестве ДНК для доставки, ДНК может обычно, но не обязательно, включать контрольную область, например, содержащую промотор, для осуществления экспрессии. Подходящие промоторы для последовательностей молекул Cas9 включают, например, промоторы CMV, EF-1a, EFS, MSCV, PGK или CAG. Подходящие промоторы для гРНК включают, например, промоторы H1, EF-1a, тРНК или U6. Промоторы с аналогичной или различающейся эффективностью могут быть выбраны для регуляции экспрессии компонентов. Последовательности, кодирующие молекулу Cas9, могут содержать сигнал ядерной локализации (NLS), например, NLS SV40. В одном варианте осуществления промотор для молекулы Cas9 или молекулы гРНК может быть независимо индуцируемым, тканеспецифичным или клеточно-специфичным. В некоторых вариантах осуществления агент, способный индуцировать генетическое нарушение, вводит комплексы RNP. Комплексы RNP включают последовательность рибонуклеотидов, таких как молекула РНК или гРНК, и белок, такой как белок Cas9 или его вариант. В некоторых вариантах осуществления белок Cas9 доставляется в виде рибонуклеопротеинового комплекса (RNP), который включает белок Cas9, представленный в настоящем описании, и молекулу гРНК, представленную в настоящем документе, например, гРНК, нацеленную на TRAC, TRBC1 и/или TRBC2. В некоторых вариантах осуществления RNP, который включает одну или более молекул гРНК, нацеленных на TRAC, TRBC1 и/или TRBC2, таких как любой, как описано, и фермент Cas9 или его вариант, непосредственно вводятся в клетку посредством физической доставки (например, электропорацией, генной пушки, трансфекция фосфатом кальция, сжатия или сдавливания клеток) липосом или наночастиц. В конкретных вариантах осуществления RNP включает одну или более молекул гРНК, нацеленных на TRAC, TRBC1 и/или TRBC2, и фермент Cas9 или его вариант вводят посредством электропорации.
[0844] В Таблице 19 приведены примеры формы, в которой компоненты могут быть доставлены в клетку-мишень.
Таблица 19 Иллюстративные способы доставки
[0845] В Таблице 20 суммированы различные способы доставки для компонентов системы Cas, например, компонента молекулы Cas9 и компонента молекулы гРНК, как описано в настоящем документе.
Таблица 20 Сравнение иллюстративных способов доставки
(a) ДНК-доставка молекулы Cas9 и/или молекулы гРНК
[0846] ДНК, кодирующая молекулы Cas9 (например, молекулы eaCas9) и/или молекулы гРНК, может быть доставлена в клетки известными в данной области способами или как описано в настоящем документе. Например, ДНК, кодирующая Cas9 и/или кодирующую gRNA, может быть доставлена, например, векторами (например, вирусными или невирусными векторами), не векторными методами (например, с использованием голой ДНК или комплексов ДНК) или их комбинацией.
[0847] В некоторых вариантах осуществления ДНК, кодирующая Cas9 и/или гРНК, доставляется вектором (например, вирусным вектором/вирусом или плазмидой).
[0848] Вектор может содержать последовательность, которая кодирует молекулу Cas9 и/или молекулу гРНК. Вектор также может содержать последовательность, кодирующую сигнальный пептид (например, для ядерной локализации, нуклеолярной локализации, митохондриальной локализации), слитый, например, с последовательностью молекулы Cas9. Например, вектор может содержать последовательность ядерной локализации (например, из SV40), слитую с последовательностью, кодирующей молекулу Cas9.
[0849] Один или более регуляторных/контрольных элементов, например, промотор, энхансер, интрон, сигнал полиаденилирования, консенсусная последовательность Козака, сайты внутренней посадки рибосомы (IRES), последовательность 2А и акцептор или донор сплайсинга могут быть включены в векторы. В одном варианте осуществления промотор распознается РНК-полимеразой II (например, промотор CMV). В другом варианте осуществления промотор распознается РНК-полимеразой III (например, промотор U6). В другом варианте осуществления промотор представляет собой регулируемый промотор (например, индуцируемый промотор). В другом варианте осуществления промотор является конститутивным промотором. В другом варианте осуществления промотор представляет собой тканеспецифичный промотор. В другом варианте осуществления промотор представляет собой вирусный промотор. В другом варианте осуществления промотор представляет собой невирусный промотор.
[0850] В одном варианте осуществления вектор или носитель доставки представляет собой вирусный вектор (например, для генерации рекомбинантных вирусов). В одном варианте осуществления вирус представляет собой ДНК-вирус (например, вирус dsДНК или ssДНК). В одном варианте осуществления вирус представляет собой РНК-вирус (например, вирус ssРНК). Иллюстративные вирусные векторы/вирусы включают, например, ретровирусы, лентивирусы, аденовирус, аденоассоциированный вирус (AAV), вирусы коровьей оспы, поксвирусы и вирусы простого герпеса.
[0851] В одном варианте осуществления вирус инфицирует делящиеся клетки. В другом варианте осуществления вирус инфицирует неделящиеся клетки. В другом варианте осуществления вирус инфицирует как делящиеся, так и неделящиеся клетки. В другом варианте осуществления вирус может интегрироваться в геном хозяина. В другом варианте осуществления вирус сконструирован так, чтобы иметь пониженный иммунитет, например, у человека. В другом варианте осуществления вирус способен к репликации. В другом варианте осуществления вирус является дефектным по репликации, например, имея делецию или замену на другие гены одной или более кодирующих областей для генов, необходимых для дополнительных циклов репликации и/или упаковки вириона. В другом варианте осуществления вирус вызывает транзиторную экспрессию молекулы Cas9 и/или молекулы гРНК. В другом варианте осуществления вирус вызывает продолжительную экспрессию молекулы Cas9, например, по меньшей мере, 1 неделю, 2 недели, 1 месяц, 2 месяца, 3 месяца, 6 месяцев, 9 месяцев, 1 год, 2 года или постоянную экспрессию молекулы Cas9 и/или молекулы гРНК. Способность упаковки вирусов может варьироваться, например, от, по меньшей мере, примерно 4 т.п.н. до, по меньшей мере, приблизительно 30 т.п.н., например, по меньшей мере, примерно 5 т.п.н., 10 т.п.н., 15 т.п.н., 15 т.п.н., 20 т.п.н., 25 т.п.н., 30 т.п.н., 35 т.п.н., 40 т.п.н., 45 т.п.н. или 50 т.п.н..
[0852] В одном варианте осуществления ДНК, кодирующая Cas9 и/или гРНК, доставляется рекомбинантным ретровирусом. В другом варианте осуществления ретровирус (например, вирус мышиного лейкоза Молони) содержит обратную транскриптазу, например, которая позволяет интегрироваться в геном хозяина. В одном варианте осуществления ретровирус является компетентным для репликации. В другом варианте осуществления ретровирус является дефектным по репликации, например, имея делецию или замену на другие гены одной или более кодирующих областей для генов, необходимых для дополнительных циклов репликации и упаковки вириона.
[0853] В одном варианте осуществления ДНК, кодирующая Cas9 и/или гРНК, доставляется рекомбинантным лентивирусом. Например, лентивирус является дефектным по репликации, например, не содержит одного или более генов, необходимых для репликации вируса.
[0854] В одном варианте осуществления ДНК, кодирующая Cas9 и/или гРНК, доставляется рекомбинантным аденовирусом. В другом варианте осуществления аденовирус сконструирован так, чтобы иметь пониженный иммунитет у человека.
[0855] В одном варианте осуществления ДНК, кодирующая Cas9 и/или гРНК, доставляется рекомбинантным AAV. В варианте осуществления AAV может включать свой геном в геном клетки-хозяина, например клетки-мишени, как описано в настоящем документе. В другом варианте осуществления AAV представляет собой самокомплементарный аденоассоциированный вирус (scAAV), например scAAV, который упаковывает обе цепи, которые отжигаются вместе с образованием двухцепочечной ДНК. Серотипы AAV, которые можно использовать в раскрытых способах, включают AAV1, AAV2, модифицированный AAV2 (например, модификации в Y444F, Y500F, Y730F и/или S662V), AAV3, модифицированный AAV3 (например, модификации в Y705F, Y731F и/или T492V).), AAV4, AAV5, AAV6, модифицированный AAV6 (например, модификации в S663V и/или T492V), AAV8, AAV 8.2, AAV9, AAV rh l0 и псевдотипированный AAV, такой как AAV2/8, AAV2/5 и AAV2/6, также могут быть использованы в раскрытых способах.
[0856] В одном варианте осуществления ДНК, кодирующая Cas9 и/или гРНК, доставляется гибридным вирусом, например гибридом одного или более вирусов, описанных в настоящем документе.
[0857] Упаковочная клетка используется для образования вирусной частицы, способной инфицировать клетку-мишень. Такая клетка включает клетку 293, которая может упаковывать аденовирус, и клетку ψ2 или клетку PA317, которая может упаковывать ретровирус. Вирусный вектор, используемый в генной терапии, обычно генерируется линией клеток-продуцентов, которые упаковывают вектор нуклеиновой кислоты в вирусную частицу. Вектор обычно содержит минимальные вирусные последовательности, необходимые для упаковки и последующей интеграции в клетку-хозяин или клетку-мишень (если применимо), при этом другие вирусные последовательности заменяются экспрессирующей кассетой, кодирующей подлежащий экспрессии белок, например, Cas9. Например, вектор AAV, используемый в генной терапии, обычно содержит только последовательности инвертированного концевого повтора (ITR) из генома AAV, которые необходимы для упаковки и экспрессии гена в клетке-хозяине или клетке-мишени. Отсутствующие вирусные функции обеспечиваются в транс-активной упаковочной клеточной линии. С этого момента вирусная ДНК упакована в клеточную линию, которая содержит плазмиду-помощник, кодирующую другие гены AAV, а именно rep и cap, но без последовательностей ITR. Клеточная линия также инфицируется аденовирусом в качестве помощника. Вирус-помощник способствует репликации вектора AAV и экспрессии генов AAV из плазмиды-помощника. Плазмида-помощник не упаковывается в значительных количествах из-за отсутствия последовательностей ITR. Загрязнение аденовирусом может быть уменьшено, например, тепловой обработкой, к которой аденовирус более чувствителен, чем AAV.
[0858] В одном варианте осуществления вирусный вектор обладает способностью распознавания типа клеток. Например, вирусный вектор может быть псевдотипирован с другим/альтернативным вирусным оболочечным гликопротеином; сконструирован со специфическим для типа клеток рецептором (например, генетическая модификация гликопротеинов вирусной оболочки для включения нацеливающих лигандов, таких как пептидный лиганд, одноцепочечное антитело, фактор роста); и/или сконструирован так, чтобы иметь молекулярный мостик с двойной специфичностью, при этом один конец распознает вирусный гликопротеин, а другой конец распознает фрагмент поверхности клетки-мишени (например, лиганд-рецептор, моноклональное антитело, авидин-биотин и химическую конъюгацию).
[0859] В одном варианте осуществления вирусный вектор достигает экспрессии, специфической для типа клеток. Например, тканеспецифичный промотор может быть сконструирован для ограничения экспрессии трансгена (Cas 9 и гРНК) только в конкретной клетке-мишени. Специфичность вектора также может быть опосредована микроРНК-зависимым контролем экспрессии трансгена. В одном варианте осуществления вирусный вектор обладает повышенной эффективностью слияния вирусного вектора и мембраны клетки-мишени. Например, слитый белок, такой как способный к слиянию гемагглютинин (HA), может быть включен для увеличения поглощения вируса клетками. В одном варианте осуществления вирусный вектор обладает способностью ядерной локализации. Например, вирус, который требует разрушения ядерной мембраны (во время клеточного деления) и, следовательно, не будет инфицировать неделящуюся клетку, может быть изменен для включения пептида ядерной локализации в матричный белок вируса, тем самым обеспечивая возможность трансдукции пролиферирующих клеток.
[0860] В одном варианте осуществления ДНК, кодирующая Cas9 и/или гРНК, доставляется не векторным способом (например, с использованием голой ДНК или комплексов ДНК). Например, ДНК может быть доставлена, например, органически модифицированным кремнеземом или силикатом (Ormosil), электропорацией, транзиторным сжатием или сдавливанием клеток (например, как описано в Lee et al. [2012] Nano Lett 12: 6322-27), генной пушкой, сонопорацией, магнитофекцией, липид-опосредованная трансфекцией, дендримерами, неорганическими наночастицами, фосфатом кальция или их комбинацией.
[0861] В одном варианте осуществления доставка посредством электропорации включает смешивание клеток с ДНК, кодирующей Cas9 и/или гРНК, в картридже, камере или кювете и подачу одного или более электрических импульсов определенной длительности и амплитуды. В одном варианте осуществления доставка посредством электропорации осуществляется с использованием системы, в которой клетки смешиваются с ДНК, кодирующей Cas9 и/или gRNA, в сосуде, соединенном с устройством (например, насосом), который подает смесь в картридж, камеру или кювету, в которой подается один или более электрических импульсов определенной длительности и амплитуды, после чего клетки доставляются во второй сосуд.
[0862] В одном варианте осуществления ДНК, кодирующая Cas9 и/или гРНК, доставляется комбинацией векторного и не векторного метода. Например, виросома содержит липосому, объединенную с инактивированным вирусом (например, вирусом HIV или вирусом гриппа), который может привести к более эффективному переносу гена, чем только вирусный или липосомный метод.
[0863] В одном варианте осуществления носитель доставки представляет собой невирусный вектор. В одном варианте осуществления невирусный вектор представляет собой неорганическую наночастицу. Иллюстративные неорганические наночастицы включают, например, магнитные наночастицы (например, Fe3MnO2) и диоксид кремния. Наружная поверхность наночастицы может быть конъюгирована с положительно заряженным полимером (например, полиэтиленимином, полилизином, полисерином), который позволяет прикреплять (например, конъюгировать или захватывать) полезную нагрузку. В одном варианте осуществления невирусный вектор представляет собой органическую наночастицу. Иллюстративные органические наночастицы включают, например, липосомы SNALP, которые содержат катионные липиды вместе с нейтральными липидами-помощниками, которые покрыты полиэтиленгликолем (ПЭГ), и комплексы протамин-нуклеиновая кислота, покрытые липидом.
[0864] Иллюстративные липиды для переноса генов представлены ниже в Таблице 21.
Таблица 21 Липиды, используемые для переноса генов
[0865] Иллюстративные полимеры для переноса генов прдставлены ниже в Таблице 22.
Таблица 22 Полимеры, используемые для переноса генов
[0866] В одном варианте осуществления носитель имеет нацеливающие модификации для увеличения поглощения клеток-мишеней наночастиц и липосом, например, клеточно-специфических антигенов, моноклональных антител, одноцепочечных антител, аптамеров, полимеров, сахаров и пептидов, проникающих в клетки. В одном варианте осуществления носитель использует фузогенные и дестабилизирующие эндосомы пептиды/полимеры. В варианте осуществления носитель претерпевает вызванные кислотой конформационные изменения (например, чтобы ускорить эндосомное освобождение карго). В одном варианте осуществления расщепляемый стимулом полимер используется, например, для высвобождения в клеточном компартменте. Например, могут быть использованы катионные полимеры на основе дисульфида, которые расщепляются в восстановливающих условиях клеточной среды.
[0867] В варианте осуществления носитель для доставки представляет собой биологический невирусный носитель для доставки. В одном варианте осуществления носитель представляет собой аттенуированную бактерию (например, природную или искусственно сконструированную, чтобы быть инвазивной, но аттенуированной для предотвращения патогенеза и экспрессии трансгена (например, Listeria monocytogenes, некоторые штаммы Salmonella, Bifidobacterium longum и модифицированная Escherichia coli), бактерии, имеющие питательный и тканеспецифичный тропизм к определенным клеткам (бактерии, имеющие модифицированные поверхностные белки для изменения специфичности клеток-мишеней). В одном варианте осуществления носитель представляет собой генетически модифицированный бактериофаг (например, сконструированные фаги, обладающие большой упаковочной способностью, меньшей иммуногенностью, содержащие последовательности поддержания плазмиды млекопитающих и включающие нацеливающие лиганды). В одном варианте осуществления носитель представляет собой вирусоподобную частицу млекопитающего. Например, могут быть получены модифицированные вирусные частицы (например, путем очистки «пустых» частиц с последующей сборкой вируса ex vivo с желаемым карго). Носитель также может быть сконструирован так, чтобы включать нацеливающие лиганды для изменения специфичности ткани мишени. В одном варианте осуществления носитель представляет собой биологическую липосому. Например, биологическая липосома представляет собой частицу на основе фосфолипида, полученную из клеток человека (например, фиктивных эритроцитов, которые представляют собой эритроциты, расщепленные на сферические структуры, полученные от пациента (например, нацеливание на ткань может быть достигнуто путем прикрепления различных ткане- или клеточно-специфических лигандов), или секреторные экзосомы - связанные с мембраной нановезикулы (30-100 нм) эндоцитозного происхождения (например, могут быть получены из клеток различных типов и поэтому могут поглощаться клетками без необходимости нацеливания на лиганды).
[0868] В одном варианте осуществления доставляются одна или более молекул нуклеиновой кислоты (например, молекулы ДНК), отличных от компонентов системы Cas, например компонента молекулы Cas9 и/или компонента молекулы гРНК, описанных в настоящем документе. В одном варианте осуществления молекула нуклеиновой кислоты доставляется одновременно с одним или более компонентами системы Cas. В одном варианте осуществления молекула нуклеиновой кислоты доставляется до или после (например, с интервалом менее чем примерно 30 минут, 1 час, 2 часа, 3 часа, 6 часов, 9 часов, 12 часов, 1 день, 2 дня, 3 дня, 1 неделю, 2 недели или 4 недели) одного или более компонентов системы Cas. В одном варианте осуществления молекула нуклеиновой кислоты доставляется способом, отличным от способа доставки одного или более компонентов системы Cas, например компонента молекулы Cas9 и/или компонента молекулы гРНК. Молекула нуклеиновой кислоты может быть доставлена любым из способов доставки, описанных в настоящем документе. Например, молекула нуклеиновой кислоты может доставляться вирусным вектором, например ретровирусом или лентивирусом, а компонент молекулы Cas9 и/или компонент молекулы гРНК может доставляться электропорацией. В одном варианте осуществления молекула нуклеиновой кислоты кодирует ген TRAC, ген TRBC или ген CAR.
(b) Доставка РНК, кодирующей молекулу Cas9
[0869] РНК, кодирующая молекулы Cas9 (например, молекулы eaCas9, молекулы eiCas9 или слитые белки eiCas9) и/или молекулы гРНК, могут быть доставлены в клетки, например клетки-мишени, описанные в настоящем документе, известными в данной области способами или как описано в настоящем документе. Например, Cas9-кодирующая и/или гРНК-кодирующая РНК может быть доставлена, например, путем микроинъекции, электропорации, транзиторного сжатия или сдавливания клеток (например, как описано в Lee et al. [2012] Nano Lett 12: 6322-27) липид-опосредованной трансфекции, пептид-опосредованной доставки или их комбинации.
[0870] В одном варианте осуществления доставка посредством электропорации включает смешивание клеток с РНК, кодирующей молекулы Cas9 (например, молекулы eaCas9, молекулы eiCas9 или слитые белки eiCas9) и/или молекулы гРНК, в картридже, камере или кювете и подачу одного или более электрических импульсов определенной продолжительности и амплитуды. В одном варианте осуществления доставка посредством электропорации осуществляется с использованием системы, в которой клетки смешиваются с РНК, кодирующей молекулы Cas9 (например, молекулы eaCas9, молекулы eiCas9 или слитые белки eiCas9) и/или молекулы гРНК в сосуде, подключенном к устройству (например, насосу), который подает смесь в картридж, камеру или кювету, в которую подается один или более электрических импульсов определенной длительности и амплитуды, после чего клетки доставляются во второй сосуд.
(c) Доставка белка Cas9 и рибонуклеопротеина (RNP)
[0871] В некоторых вариантах осуществления один или более агентов, способных вводить расщепление, например система Cas9/гРНК, вводятся в клетку в виде рибонуклеопротеинового комплекса (RNP). Комплексы RNP включают последовательность рибонуклеотидов, таких как молекула РНК или гРНК, и белок, такой как белок Cas9 или его вариант. Например, белок Cas9 доставляется в виде комплекса RNP, который включает белок Cas9 и молекулу гРНК, нацеленную на последовательность-мишень, например, с помощью электропорации или другого метода физической доставки. В некоторых вариантах осуществления RNP доставляется в клетку посредством электропорации или другими физическими средствами, например, с помощью генной пушки, трансфекции фосфатом кальция, сжатия или сдавливания клетки. В некоторых вариантах осуществления RNP может пересекать плазматическую мембрану клетки без необходимости в дополнительных агентах доставки (например, низкомолекулярных агентах, липидах и т. д.).
[0872] В некоторых вариантах осуществления доставка одного или более агентов, способных индуцировать генетическое нарушение, например CRISPR/Cas9, в качестве RNP дает преимущество в том, что направленное нарушение происходит транзиторно, например, в клетках, в которые введен RNP, без размножения агента в потомстве клеток. Например, доставка с помощью RNP сводит к минимуму наследование агента потомством клеток, тем самым уменьшая вероятность ошибочного генетического нарушения в потомстве.
[0873] В некоторых вариантах осуществления более чем один агент(ы) или его компоненты доставляются в клетку. Например, в некоторых вариантах осуществления агент(ы), способный вызывать генетическое нарушение двух или более участков в геноме, например, локусов TRAC, TRBC1 и/или TRBC2, доставляется в клетку. В некоторых вариантах осуществления агент(ы) и их компоненты доставляются с использованием одного способа. Например, в некоторых вариантах осуществления агент(ы) для индукции генетического нарушения локусов TRAC, TRBC1 и/или TRBC2 доставляются в виде полинуклеотидов, кодирующих компоненты для генетического нарушения. В некоторых вариантах осуществления один полинуклеотид может кодировать агенты, которые нацелены на локусы TRAC, TRBC1 и/или TRBC2. В некоторых вариантах осуществления два или более разных полинуклеотида могут кодировать агенты, которые нацелены на локусы TRAC, TRBC1 и/или TRBC2. В некоторых вариантах осуществления агенты, способные индуцировать генетическое нарушение, могут доставляться в виде комплексов рибонуклеопротеинов (RNP), и два или более различных комплексов RNP могут доставляться вместе в виде смеси или по отдельности.
[0874] Молекулы Cas9 (например, молекулы eaCas9, молекулы eiCas9 или слитые белки eiCas9) могут быть доставлены в клетки известными в данной области способами или как описано в настоящем документе. Например, белковые молекулы Cas9 могут быть доставлены, например, путем микроинъекции, электропорации, транзиторного сжатия или сдавливания клеток (например, как описано в Lee et al. [2012] Nano Lett 12: 6322-27), липид-опосредованной трансфекции, пептид-опосредованной доставки или их комбинации. Доставка может сопровождаться ДНК, кодирующей гРНК или с помощью гРНК. В некоторых вариантах осуществления белок Cas9 доставляется в виде рибонуклеопротеинового комплекса (RNP), который включает белок Cas9, представленный в настоящем описании, и молекулу гРНК, представленную в настоящем документе, например, гРНК, нацеленную на TRAC, TRBC1 и/или TRBC2. В некоторых вариантах осуществления комплекс RNP включает последовательность рибонуклеотидов, такую как молекула РНК или гРНК, и белок, такой как белок Cas9 или его вариант. В некоторых вариантах осуществления RNP, который включает одну или более молекул гРНК, нацеленных на TRAC, TRBC1 и/или TRBC2, таких как любой описанный, и фермент Cas9 или его вариант, непосредственно вводится в клетку посредством физической доставки (например, электропорации, генной пушки, трансфекции фосфатом кальция, сжатия или сдавливания клеток) липосом или наночастиц. В конкретных вариантах осуществления RNP включает одну или более молекул гРНК, нацеленных на TRAC, TRBC1 и/или TRBC2, таких как любые описанные, и фермент Cas9 или его вариант вводят посредством электропорации.
[0875] В одном варианте осуществления доставка посредством электропорации включает смешивание клеток с молекулами Cas9 (например, молекулами eaCas9, молекулами eiCas9 или слитыми белками eiCas9) с или без молекул гРНК в картридже, камере или кювете и подачу одного или более электрических импульсов определенной длительности и амплитуды. В одном варианте осуществления доставка посредством электропорации осуществляется с использованием системы, в которой клетки смешаны с молекулами Cas9 (например, молекулами eaCas9, молекулами eiCas9 или белками слияния eiCas9) с молекулами гРНК или без них в сосуде, подключенном к устройству (например, насосу), который подает смесь в картридж, камеру или кювету, в которую подается один или более электрических импульсов определенной длительности и амплитуды, после чего клетки доставляются во второй сосуд.
B. Направленная интеграция с помощью направляемой гомологией репарации (HDR)
[0876] В некоторых вариантах осуществления, предложенных в настоящем описании, направляемая гомологией репарация (HDR) может использоваться для направленной интеграции конкретной части матричного полинуклеотида, содержащего трансген, например последовательность нуклеиновой кислоты, кодирующую любой из представленных рекомбинантных рецепторов, например рекомбинантный T клеточный рецептор (TCR), в определенный участок в геноме, например, локус TRAC, TRBC1 и/или TRBC2. В некоторых вариантах осуществления матричный полинуклеотид, содержащий последовательность нуклеиновой кислоты, например трансген, кодирующий рекомбинантный Т-клеточный рецептор (TCR) или его антигенсвязывающий фрагмент или цепь, вводится в клетку, например иммунную клетку, имеющую генетическое нарушение сайта-мишени в гене константной области Т-клеточного рецептора альфа (TRAC) и/или в гене константной области Т-клеточного рецептора бета (TRBC). В некоторых вариантах осуществления последовательность нуклеиновой кислоты или трансген, кодирующий рекомбинантный TCR или его антигенсвязывающий фрагмент или его цепь, направляются для интеграции в сайт-мишень или рядом с ним посредством направляемой гомологией репарации (HDR). В конкретных вариантах осуществления интеграция в сайт-мишень или рядом с ним происходит в пределах части кодирующей последовательности гена TRAC и/или TRBC, такой как, например, часть кодирующей последовательности ниже или в 3'-области от сайта-мишени.
[0877] В некоторых вариантах осуществления сайт-мишень находится в гене константной области Т-клеточного рецептора альфа (TRAC). В некоторых вариантах осуществления сайт(сайты)-мишень находится в гене константной области 1 Т-клеточного рецептора бета (TRBC1) или константной области 2 Т-клеточного рецептора бета (TRBC2). В некоторых вариантах осуществления один или более сайтов-мишеней находятся в гене TRAC и один или оба из гена TRBC1 и TRBC2. В некоторых вариантах осуществления матричный полинуклеотид, содержащий последовательность нуклеиновой кислоты и/или трансген, кодирующий рекомбинантный рецептор, такой как любой из представленных TCR или его часть, вводится в иммунную клетку, имеющую генетическое нарушение одного или более сайтов-мишеней в пределах TRAC, TRBC1 и/или TRBC2, направленное на один сайт-мишень из, по меньшей мере, одного сайта-мишени посредством HDR, или рядом с ним.
[0878] В некоторых вариантах осуществления направленное генетическое нарушение и направленная интеграция нуклеиновых кислот, кодирующих рекомбинантный рецептор, посредством HDR происходит в одном или более сайтах-мишенях (также известных как «положение-мишень», «последовательность ДНК-мишени» или «локализация-мишень») эндогенных генов, которые кодируют один или более доменов, областей и/или цепей эндогенного Т-клеточного рецептора (TCR). В некоторых вариантах осуществления варианты осуществления, представленные в настоящем описании, включают одно или более направленных генетических нарушений, например разрыв ДНК, в одном или более локусах эндогенного гена TCR (как например, эндогенные гены, кодирующие константные домены TCRα и/или TCRβ) методами редактирования генов в комбинации с направленным нокином нуклеиновых кислот, кодирующих рекомбинантный рецептор (как например, рекомбинантный TCR или CAR), путем направляемой гомологией репарации (HDR). В некоторых вариантах осуществления разрыв ДНК происходит в результате стадии редактирования генов, например разрывов ДНК, генерируемых направленными нуклеазами, используемыми для введения направленного генетического нарушения, такого как любые описанные здесь. Примеры способов редактирования генов эндогенных локусов TCR известны и включают, но не ограничиваются ими, описанные в настоящем документе или в другом месте, например, в публикациях США № US 2011/0158957, US 2014/0301990, US 2015/0098954, US2016/0208243, US2016/272999 и US2015/056705; Международных публикациях PCT №. WO2014/191128, WO2015/136001, WO2015/161276, WO2016/069283, WO2016/016341, WO2017/193107 и WO2017/093969; и в Osborn et al. (2016) Mol. Ther. 24 (3): 570-581.
[0879] Изменение последовательностей нуклеиновых кислот в сайте-мишени может происходить с помощью HDR с экзогенно предоставленным матричным полинуклеотидом (также называемым донорным полинуклеотидом или матричной последовательностью). Например, матричный полинуклеотид обеспечивает изменение последовательности-мишени, такое как вставка трансгена, содержащегося в матричном полинуклеотиде. В некоторых вариантах осуществления плазмиду или вектор можно использовать в качестве матрицы для гомологичной рекомбинации. В некоторых вариантах осуществления линейный фрагмент ДНК можно использовать в качестве матрицы для гомологичной рекомбинации. В некоторых вариантах осуществления одноцепочечный матричный полинуклеотид можно использовать в качестве матрицы для изменения последовательности-мишени альтернативными методами направляемой гомологией репарации (например, отжиг одной цепи) между последовательностью-мишенью и матричным полинуклеотидом. Модифицированное полинуклеотидом изменение последовательности-мишени зависит от расщепления нуклеазой, например направленной нуклеазой, такой как CRISPR/Cas9. Расщепление нуклеазой может включать двухцепочечный разрыв или два одноцепочечных разрыва.
[0880] В некоторых вариантах осуществления способы для HDR включают введение в иммунную клетку, например, Т-клетку, одного или более агентов, где каждый из одного или более агентов независимо способен индуцировать генетическое нарушение гена константной области Т-клеточного рецептора альфа (TRAC) и/или гена константной области Т-клеточного рецептора бета (TRBC). В некоторых вариантах осуществления один или более агентов могут включать нуклеазу цинкового пальца (ZFN), TAL-эффекторную нуклеазу (TALEN) или комбинацию CRISPR-Cas9, которая специфически связывается, распознает или гибридизуется с сайтом-мишенью в локусе TRAC или TRBC, например, с использованием методов, описанных выше в разделе V.A. В некоторых вариантах осуществления один или более агентов вводят, используя комбинацию CRISPR-Cas9, в которой каждый из одного или более агентов содержит гид-РНК (гРНК), содержащую нацеливающий домен, который комплементарен по меньшей мере одному сайту-мишени. Любые последовательности нацеливающих доменов гРНК, которые нацелены на локус TRAC или TRBC, можно использовать для осуществления генетического нарушения, включая любые, описанные выше. В некоторых вариантах осуществления локус TRAC для нацеливания на локус TRAC представляет собой GAGAAUCAAAAUCGGUGAAU (SEQ ID NO: 1048). В некоторых вариантах осуществления последовательность нацеливающего домена гРНК для нацеливания на локус TRBC представляет собой GGCCUCGGCGCUGACGAUCU (SEQ ID NO: 1053).
[0881] В некоторых вариантах осуществления HDR включает введение в иммунную клетку, например T-клетку, одной или более нацеливающих последовательностей гРНК, способных индуцировать генетическое нарушение гена константной области T-клеточного рецептора альфа (TRAC).
[0882] В некоторых вариантах осуществления генетическое нарушение осуществляют путем введения в клетку последовательности нацеливающего домена гРНК вместе с Cas9, например, с использованием способов, описанных выше. Cas9 может быть введен в виде нуклеиновой кислоты или в виде кодируемого белка. В некоторых вариантах осуществления генетическое нарушение осуществляют путем введения в клетку, например посредством электропорации, комплекса рибонуклеопротеина (RNP), содержащего гРНК и белок Cas9, например, с использованием способов, описанных выше.
В некоторых вариантах осуществления способы HDR дополнительно включают введение в клетку полинуклеотида, такого как матричный полинуклеотид, который содержит (а) последовательность нуклеиновой кислоты, кодирующую любую из предложенных TCR или его антигенсвязывающих частей, и (b) одно или более плечей гомологии, связанных с последовательностью нуклеиновой кислоты, где одно или более плеч гомологии содержат последовательность, гомологичную одной или более областям открытой рамки считывания локуса константной области Т-клеточного рецептора альфа (TRAC). В некоторых вариантах осуществления одно или более плеч гомологии облегчают передачу генетической информации из матричного полинуклеотида в локус-мишень, например, сайт-мишень локуса TRAC.
[0883] В некоторых вариантах осуществления «рекомбинация» относится к процессу обмена генетической информацией между двумя полинуклеотидами. В некоторых вариантах осуществления «гомологичная рекомбинация (HR)» относится к специализированной форме такого обмена, который имеет место, например, во время репарации двухцепочечных разрывов в клетках с помощью механизмов репарации, направляемой гомологией. Этот процесс требует гомологии нуклеотидной последовательности, использует матричный полинуклеотид для матричной репарации ДНК-мишени (то есть той, которая подверглась двухцепочечному разрыву, например, сайт-мишень в эндогенном гене) и по-разному известен как «не-кроссоверная генная конверсия» или«генная конверсия короткого тракта», потому что это приводит к переносу генетической информации от матричного полинуклеотида к мишени. В некоторых вариантах осуществления такой перенос может включать коррекцию ошибочного спаривания гетеродуплексной ДНК, которая образуется между поврежденной мишенью, например, из-за генетического нарушения, и матричным полинуклеотидом, и/или «отжиг цепи в зависимости от синтеза», в котором матричный полинуклеотид используется для повторного синтеза генетической информации, которая станет частью мишени, и/или связанных процессов. Такая специализированная HR часто приводит к изменению последовательности молекулы-мишени, так что часть или вся последовательность матричного полинуклеотида включается в полинуклеотид-мишень.
[0884] В некоторых вариантах осуществления матричный полинуклеотид, например полинуклеотид, содержащий трансген, интегрируется в геном клетки с помощью механизмов, независимых от гомологии. Способы включают создание двухцепочечного разрыва (DSB) в геноме клетки и расщепление матричной полинуклеотидной молекулы с использованием нуклеазы, так что матричный полинуклеотид интегрируется в сайт DSB. В некоторых вариантах осуществления матричный полинуклеотид интегрируется не зависящими от гомологии способами (например, NHEJ). После in vivo-расщепления матричные полинуклеотиды могут быть целенаправленно интегрированы в геном клетки в место расположения DSB. Матричный полинуклеотид может включать один или более одинаковых сайтов-мишеней для одной или более нуклеаз, используемых для создания DSB. Таким образом, матричный полинуклеотид может быть расщеплен одной или болееи одинаковыми нуклеазами, используемыми для расщепления эндогенного гена, в котором желательна интеграция. В некоторых вариантах осуществления матричный полинуклеотид включает сайты-мишени нуклеаз, отличные от нуклеаз, используемых для индукции DSB. Как описано в настоящем документе, генетическое нарушение сайта-мишени или положения-мишени может быть создано любыми механизмами, такими как ZFN, TALEN, система CRISPR/Cas9 или нуклеазы TtAgo.
[0885] В некоторых вариантах осуществления, предложенных в настоящем описании, направляемая гомологией репарация (HDR) может использоваться для направленной интеграции конкретной части матричного полинуклеотида, содержащего трансген, например последовательность нуклеиновой кислоты, кодирующую любой из представленных рекомбинантных рецепторов, например рекомбинантные TCR, в определенный участок в геноме, например, локус TRAC, TRBC1 и/или TRBC2. В некоторых вариантах осуществления наличие генетического нарушения (например, разрыва ДНК) и полинуклеотида, например, матричного полинуклеотида, содержащего одно или более плеч гомологии (например, последовательности нуклеиновой кислоты, гомологичные последовательностям, окружающим генетическое нарушение), используются для индукции или прямой HDR с гомологичными последовательностями, действующими в качестве матрицы для репарации ДНК.
[0886] В некоторых вариантах осуществления матричный полинуклеотид, имеющий гомологию с последовательностями в одном или более сайтах-мишенях в эндогенной ДНК или рядом с ними, можно использовать для изменения структуры ДНК-мишени, например, направленной вставки трансгена. В некоторых вариантах осуществления матричный полинуклеотид содержит последовательности гомологии (например, плечи гомологии), фланкирующие трансген, например последовательности нуклеиновой кислоты, кодирующие рекомбинантный рецептор, для направленной вставки. В некоторых вариантах осуществления гомологичные последовательности нацелены на трансген в одном или более из локусов TRAC, TRBC1 и/или TRBC2. В некоторых вариантах осуществления матричный полинуклеотид включает дополнительные последовательности (кодирующие или некодирующие последовательности) между плечами гомологии, такие как регуляторные последовательности, такие как промоторы и/или энхансеры, донорные и/или акцепторные сайты сплайсинга, сайт внутренней посадки рибосомы (IRES), последовательности, кодирующие элементы пропуска рибосомы (например, 2A-пептиды), маркеры и/или сайты SA, и/или один или более дополнительных трансгенов.
[0887] В некоторых вариантах осуществления матричный полинуклеотид включает или содержит трансген, часть трансгена и/или нуклеиновую кислоту, кодирующую рекомбинантный рецептор, такой как рекомбинантный TCR или его цепь, которые содержат один или более вариабельных доменов и один или более константных доменов. В некоторых вариантах осуществления рекомбинантный TCR или его цепь содержат один или более константных доменов, которые имеют полную, например, 100% или примерно 100% идентичности со всем или с частью и/или фрагментом константного домена эндогенного TCR. В некоторых вариантах осуществления трансген кодирует весь или часть константного домена, например часть или фрагмент константного домена, который полностью или частично идентичен константному домену эндогенного TCR. В некоторых вариантах осуществления трансген содержит нуклеотиды последовательности, имеющей или имеющей по меньшей мере 70, 75, 80, 85, 90, 95, 97, 98, 99, 99,5 или 99,9% идентичности со всей или с частью последовательности нуклеиновой кислоты, представленной в SEQ ID NO: 348, 349 или 1047.
[0888] В некоторых вариантах осуществления трансген содержит последовательность, кодирующую цепь TCRα и/или TCRβ или их часть, которая была оптимизирована по кодонам. В некоторых вариантах осуществления трансген кодирует часть цепи TCRα и/или TCRβ с менее чем 100% идентичностью аминокислотной последовательности с соответствующей частью нативной или эндогенной цепи TCRα и/или TCRβ. В некоторых вариантах осуществления кодируемая цепь TCRα и/или TCRβ содержит аминокислотную последовательность, которая имеет, имеет примерно или имеет по меньшей мере 70%, 75%, 80%, 85%, 90%, 95%, 98%, 99%, или более чем 99% идентичности, но менее 100% идентичности с соответствующей нативной или эндогенной цепью TCRα и/или TCRβ. В конкретных вариантах осуществления трансген кодирует константный домен TCRα и/или TCRβ или его часть с менее чем 100% идентичностью аминокислотной последовательности с соответствующим нативным или эндогенным константным доменом TCRα и/или TCRβ. В некоторых вариантах осуществления константный домен TCRα и/или TCRβ содержит аминокислотную последовательность, которая имеет, имеет примерно или имеет по меньшей мере 70%, 75%, 80%, 85%, 90%, 95%, 98%, 99%, или более чем 99% идентичности, но менее 100% идентичности с соответствующей нативной или эндогенной цепью TCRα и/или TCRβ. В конкретных вариантах осуществления трансген кодирует константные домены цепей TCRα и/или TCRβ и/или константные домены TCRα и/или TCRβ, содержащие одну или более модификаций для введения одной или более дисульфидных связей. В некоторых вариантах осуществления трансген кодирует цепь TCRα и/или TCRβ и/или TCRα и/или TCRβ с одной или болееи модификациями для удаления или предотвращения нативной дисульфидной связи, например, между TCRα, кодируемой трансгеном, и эндогенной цепью TCRβ, или между TCR β, кодируемой трансгеном, и эндогенной цепью TCR α. В некоторых вариантах осуществления один или более нативных цистеинов, которые образуют и/или способны образовывать нативную межцепочечную дисульфидную связь, замещены другим остатком, например серином или аланином. В некоторых вариантах осуществления цепи TCRα и/или TCRβ и/или константные домены цепи TCRα и/или TCRβ модифицированы для замены одного или более нецистеиновых остатков на цистеин. В некоторых вариантах осуществления один или более ненативных остатков цистеина способны образовывать ненативные дисульфидные связи, например, между рекомбинантной цепью TCRα и цепью TCRβ, кодируемой трансгеном. В некоторых вариантах осуществления цистеин вводится в один или более из остатков Thr48, Thr45, Tyr10, Thr45 и Ser15 на основании нумерации константного домена TCRα, представленного в SEQ ID NO: 1352. В некоторых вариантах осуществления цистеины могут быть введены в остаток Ser57, Ser77, Ser17, Asp59 Glu15 цепи TCR β на основани нумерации цепи TCRβ, представленной в SEQ ID NO: 1353. Иллюстративные ненативные дисульфидные связи TCR описаны в опубликованных Международных РСТ № WO 2006/000830, WO 2006/037960 и Kuball et al. (2007) Blood, 109: 2331-2338.
[0889] В некоторых вариантах осуществления трансген содержит одну или более модификаций для введения одного или более остатков цистеина, которые способны образовывать одну или более ненативных дисульфидных связей между цепью TCRα и цепью TCRβ. В некоторых вариантах осуществления трансген кодирует цепь TCRα или ее часть или фрагмент, содержащий константный домен TCRα, содержащий цистеин в положении, соответствующем положению 48 с нумерацией, представленной в SEQ ID NO: 1355. В некоторых вариантах осуществления константный домен TCRα имеет аминокислотную последовательность, представленную в любой из SEQ ID NO: 1352 или 1355, или последовательность аминокислот, которая имеет, имеет примерно или имеет, по меньшей мере, 70%, 75%, 80%. 85%, 90%, 95%, 97%, 98%, 99% идентичности последовательности с ними и содержит один или более остатков цистеина, способных образовывать ненативную дисульфидную связь с цепью TCRβ. В некоторых вариантах осуществления трансген кодирует цепь TCRβ или ее часть, содержащую константный домен TCRβ, содержащий цистеин в положении, соответствующем положению 57 с нумерацией, представленной в SEQ ID NO: 1353. В некоторых вариантах осуществления константный домен TCRα имеет аминокислотную последовательность, представленную в любой из SEQ ID NO: 1353, 1354 или 1356, или последовательность аминокислот, которая имеет, имеет примерно или имеет, по меньшей мере, 70%, 75%, 80%. 85%, 90%, 95%, 97%, 98%, 99% идентичности последовательности с ними и содержит один или более остатков цистеина, способных образовывать ненативную дисульфидную связь с цепью TCRβ.
[0890] Матричный полинуклеотид может представлять собой ДНК, одноцепочечную и/или двухцепочечную и может быть введен в клетку в линейной или циклической форме. Смотри также, Патентные публикации США № 20100047805 и 20110207221. Матричный полинуклеотид также может быть введен в форме ДНК, которая может быть введена в клетку в циклической или линейной форме. При введении в линейной форме концы матричного полинуклеотида могут быть защищены (например, от экзонуклеолитической деградации) известными способами. Например, один или более дидезоксинуклеотидных остатков добавляются к 3'-концу линейной молекулы, и/или самокомплементарные олигонуклеотиды лигируются с одним или обоими концами. Смотри, например, Chang et al. (1987) Proc. Natl. Acad. Sci. USA 84:4959-4963; Nehls et al. (1996) Science 272: 886-889. Дополнительные способы защиты экзогенных полинуклеотидов от деградации включают, но не ограничиваются ими, добавление концевой аминогруппы (аминогрупп) и использование модифицированных межнуклеотидных связей, таких как, например, фосфоротиоаты, фосфорамидаты и остатки O-метил рибозы или дезоксирибозы. При введении в двухцепочечной форме матричный полинуклеотид может включать в себя один или более нуклеазных сайтов-мишеней, например, нуклеазные сайты-мишени, фланкирующие трансген для интеграции в геном клетки. Смотри, например, Патентную публикацию № 20130326645.
[0891] В некоторых вариантах осуществления матричный полинуклеотид содержит трансгенные, например, последовательности нуклеиновой кислоты, кодирующие рекомбинантный рецептор, фланкированные гомологичными последовательностями (также называемыми «плечами гомологии») на 5'- и 3'-концах, чтобы обеспечить механизм репарации ДНК, например, механизм гомологичной рекомбинации, чтобы использовать матричный полинуклеотид в качестве матрицы для репарации, эффективно вставляя трансген в сайт-мишень интеграции в геноме. Плечо гомологии должно простираться, по меньшей мере, до области, в которой может произойти резекция конца, например, чтобы позволить разрезанному одноцепочечному выступу найти комплементарную область в матричном полинуклеотиде. В некоторых вариантах осуществления плечо гомологии не прстирается на повторяющиеся элементы, например ALU-повторы или LINE-повторы. На основе гомологии между последовательностью эндогенного гена, окружающей генетическое нарушение, и 5'- и/или 3'-плечами гомологии, включенными в полинуклеотид, например матричный полинуклеотид, механизм репарации клеточной ДНК может использовать матричный полинуклеотид для репарации разрыва ДНК и повторного синтеза генетической информации в сайте генетического нарушения, тем самым эффективно вставляя или интегрируя трансгенные последовательности в матричном полинуклеотиде в сайте генетического нарушения или рядом с ним.
[0892] В некоторых вариантах осуществления матричный полинуклеотид содержит следующие компоненты: [5′-плечо гомологии] - [трансген] - [3'-плечо гомологии]. Плечи гомологии обеспечивают рекомбинацию в хромосоме, таким образом вставляя трансген в ДНК в сайт расщепления, например, сайт-мишени (сайты-мишени) или рядом с ним. В некоторых вариантах осуществления плечи гомологии фланкируют наиболее дистальный сайт-мишень (сайты-мишени).
[0893] В некоторых аспектах трансген (например, последовательности экзогенной нуклеиновой кислоты) в матричном полинуклеотиде можно использовать для определения локализации сайтов-мишеней и/или плеч гомологии. В некоторых аспектах сайт-мишень генетического нарушения может использоваться в качестве гида для конструирования матричных полинуклеотидов и/или плеч гомологии, используемых для HDR. В некоторых вариантах осуществления генетическое нарушение может являться мишенью вблизи желаемого сайта направленной интеграции трансгенных последовательностей (например, кодирующих рекомбинантный TCR или его часть). В некоторых аспектах сайт-мишень находится в пределах экзона открытой рамки считывания локуса TRAC, TRBC1 и/или TRBC2. В некоторых аспектах сайт находится в интроне открытой рамки считывания локуса TRAC, TRBC1 и/или TRBC2.
[0894] Иллюстративные длины плеч гомологии включают по меньшей мере или по меньшей мере примерно, или составляют, или составляют примерно 50, 100, 200, 250, 300, 400, 500, 600, 700, 750, 800, 900, 1000, 2000, 3000, 4000 или 5000 нуклеотидов. Иллюстративные длины плеч гомологии включают менее чем или менее чем примерно или составляют или составляют примерно 50, 100, 200, 250, 300, 400, 500, 600, 700, 750, 800, 900, 1000, 2000, 3000, 4000 или 5000 нуклеотидов. В некоторых вариантах осуществления длина плеча гомологии составляет 50-100, 100-250, 250-500, 500-750, 750-1000, 1000-2000, 2000-3000, 3000-4000 или 4000-5000 нуклеотидов. В некоторых вариантах осуществления матричный полинуклеотид содержит, по меньшей мере, или менее или примерно 200, 300, 400, 500, 600, 700, 800, 900 или 1000 пар оснований гомологии 5'-сайта-мишени, 3'-сайта-мишени, или обоих, 5'- и 3'-сайта-мишени, например, в пределах гена, локуса или открытой рамки считывания TRAC, TRBC1 и/или TRBC2. В конкретных вариантах осуществления матричный полинуклеотид содержит нуклеотидные последовательности, например, плечи гомологии, имеющие гомологию 5'-сайта-мишени, 3'-сайта-мишени или обоих, 5'- и 3'-сайта-мишени, например, в гене, локусе или открытой рамке считывания TRAC. В некоторых вариантах осуществления пиллюстративные 5'- и 3'-плечи гомологии для направленной интеграции в локус TRAC представлены в SEQ ID NO: 1343 и 1344, соответственно.
[0895] В некоторых вариантах осуществления матричный полинуклеотид может представлять собой линейную одноцепочечную ДНК. В некоторых вариантах осуществления матричный полинуклеотид представляет собой (i) линейную одноцепочечную ДНК, которая может отжигаться на цепи ДНК-мишени, содержащей одноцепочечный разрыв (ii) линейную одноцепочечную ДНК, которая может отжигаться на интактной цепи ДНК-мишени, (iii) линейную одноцепочечную ДНК, которая может отжигаться на транскрибируемой цепи ДНК-мишени, (iv) линейную одноцепочечную ДНК, которая может отжигаться на нетранскрибированной цепи ДНК-мишени, или более одной из предшествующих.
[0896] В некоторых вариантах осуществления матричный полинуклеотид содержит плечи гомологии для нацеливания на эндогенный локус TRAC (пример нуклеотидной последовательности локуса гена TRAC человека представлен в SEQ ID NO: 348; Эталонной последовательности NCBI: NG_001332.3, TRAC или описан в Таблице 13 в настоящем документе). В некоторых вариантах осуществления генетическое нарушение локуса TRAC вводится в раннюю кодирующую область гена, включая последовательность, следующую непосредственно за стартовым сайтом транскрипции, в пределах первого экзона кодирующей последовательности или в пределах 500 п.н. от стартового сайта транскрипции (например, в пределах менее чем 500, 450, 400, 350, 300, 250, 200, 150, 100 или 50 п.н.) или в пределах 500 п.н. от старт-кодона (например, в пределах менее чем 500, 450, 400, 350, 300, 250, 200, 150, 100 или 50 п.н.). В некоторых вариантах осуществления генетическое нарушение вводят с использованием любой из направленных нуклеаз и/или гРНК. В некоторых вариантах осуществления матричный полинуклеотид содержит примерно от 500 до 1000, например, от 600 до 900 или от 700 до 800 пар оснований гомологии по обе стороны от генетического нарушения, вызванного направленными нуклеазами и/или гРНК. В некоторых вариантах осуществления матричный полинуклеотид содержит примерно 500, 600, 700, 800, 900 или 1000 пар оснований 5'-последовательностей плеч гомологии, которые гомологичны 500, 600, 700, 800, 900 или 1000 парам оснований последовательностей 5'-области генетического нарушения (например, в локусе TRAC), трансген и примерно 500, 600, 700, 800, 900 или 1000 пар оснований последовательностей 3'-области плеч гомологии, которые гомологичны 500, 600, 700, 800, 900 или 1000 парам оснований последовательностей 3'-области генетического нарушения (например, в локусе TRAC). В некоторых вариантах осуществления примеры 5'- и 3'-плечей гомологии для направленной интеграции в локус TRAC представлены в SEQ ID NO: 1343 и 1344.
[0897] В некоторых вариантах осуществления матричный полинуклеотид содержит плечи гомологии для нацеливания на эндогенный локус TRBC1 или TRBC2 (пример нуклеотидной последовательности локуса гена TRBC1 человека представлен в SEQ ID NO: 349; Эталонной последовательности NCBI: NG_001333.2, TRBC1, описанной в Таблице 14 настоящего документа; пример нуклеотидной последовательности локуса гена TRBC2 человека, представленной в SEQ ID NO: 1047; Эталонной последовательности NCBI: NG_001333.2, TRBC2, описанной в Таблице 15 настоящего документа). В некоторых вариантах осуществления генетическое нарушение локуса TRBC1 или TRBC2 вводится в раннюю кодирующую область гена, включая последовательность, непосредственно следующую за стартовым сайтом транскрипции, в пределах первого экзона кодирующей последовательности или в пределах 500 п.н. от стартового сайта транскрипции (например, в пределах менее чем 500, 450, 400, 350, 300, 250, 200, 150, 100 или 50 п.н.) или в пределах 500 п.н. от старт-кодона (например, в пределах менее чем 500, 450, 400, 350, 300, 250 п. 200, 150, 100 или 50 п.н.). В некоторых вариантах осуществления генетическое нарушение вводят с использованием любой из направленных нуклеаз и/или гРНК, описанных в настоящем документе. В некоторых вариантах осуществления матричный полинуклеотид содержит примерно от 500 до 1000, например, от 600 до 900 или от 700 до 800 пар оснований гомологии по обе стороны от генетического нарушения, вызванного направленными нуклеазами и/или гРНК. В некоторых вариантах осуществления матричный полинуклеотид содержит примерно 500, 600, 700, 800, 900 или 1000 пар оснований 5'-последовательностей плеч гомологии, которые гомологичны 500, 600, 700, 800, 900 или 1000 парам оснований последовательностей 5'-области генетического нарушения (например, в локусе TRBC1 или TRBC2), трансген и примерно 500, 600, 700, 800, 900 или 1000 пар оснований последовательностей 3'-области плеч гомологии, которые гомологичны 500, 600, 700, 800, 900 или 1000 парам оснований последовательностей 3'-области генетического нарушения (например, в локусе TRBC1 или TRBC2).
[0898] В некоторых случаях матричный полинуклеотид содержит промотор, например промотор, который является экзогенным и/или не присутствует в локусе-мишени или рядом с ним. В некоторых вариантах осуществления промотор управляет экспрессией только в конкретном типе клеток (например, промотор, специфичный для Т-клеток или В-клеток или NK-клеток). В некоторых вариантах осуществления, в которых последовательности, кодирующие функциональный полипептид, не содержат промотора, экспрессия интегрированного трансгена затем обеспечивается транскрипцией, управляемой эндогенным промотором или другим контрольным элементом в интересующей области.
[0899] Трансген, включая трансген, кодирующий рекомбинантный рецептор или его антигенсвязывающую часть, или его цепь и/или еще дополнительный трансген, может быть вставлен так, что его экспрессия управляется эндогенным промотором в сайте интеграции, а именно промотором, который управляет экспрессия эндогенного гена TCR, в который он встроен (например, TRAC, TRBC1 и/или TRBC2). Например, в некоторых вариантах осуществления кодирующие последовательности в трансгене могут быть вставлены без промотора, но в рамке с кодирующей последовательностью эндогенного гена-мишени, так что экспрессия интегрированного трансгена контролируется транскрипцией эндогенного промотора в сайте интеграции. В некоторых вариантах осуществления трансген, кодирующий рекомбинантный TCR или его антигенсвязывающий фрагмент или его цепь и/или один или более второй трансген независимо функционально связан с эндогенным промотором гена в сайте-мишени. В некоторых вариантах осуществления элемент пропуска рибосомы/элемент саморасщепления, такой как элемент 2А, расположен выше последовательности, кодирующей трансген, так что элемент пропуска рибосомы/элемент саморасщепления расположен в рамке с эндогенным геном. В некоторых вариантах осуществления трансген, кодирующий рекомбинантный TCR или его антигенсвязывающий фрагмент или его часть, функционально связан с эндогенным промотором гена в сайте-мишени (например, TRAC, TRBC1 и/или TRBC2).
[0900] В некоторых вариантах осуществления трансген может содержать промотор и/или энхансер, например конститутивный промотор или индуцируемый или тканеспецифичный промотор. В некоторых вариантах осуществления промотор представляет собой или содержит конститутивный промотор. Иллюстративные конститутивные промоторы включают, например, ранний промотор обезьяньего вируса 40 (SV40), немедленно-ранний промотор цитомегаловируса (CMV), промотор убиквитина С человека (UBC), промотор фактора элонгации 1α человека (EF1α), промотор фосфоглицераткиназы 1 мыши (PGK) и куриный промотор β-актина в сочетании с ранним энхансером CMV (CAGG). В некоторых вариантах осуществления конститутивный промотор представляет собой синтетический или модифицированный промотор. В некоторых вариантах осуществления промотор представляет собой или содержит MND-промотор, синтетический промотор, который содержит область U3 модифицированного MoMuLV LTR с энхансером вируса миелопролиферативной саркомы (последовательность представлена в SEQ ID NO: 1361 или 1347); см. Challita et al. (1995) J. Virol. 69 (2): 748-755). В некоторых вариантах осуществления промотор представляет собой тканеспецифичный промотор. В другом варианте осуществления промотор представляет собой вирусный промотор. В другом варианте осуществления промотор представляет собой невирусный промотор. В некоторых случаях промотор выбирают из промотора (EF1α) фактора элонгации человека 1 альфа (последовательность, представленная в SEQ ID NO: 1359 или 1360) или его модифицированной формы (промотор EF1α с энхансером HTLV1; последовательность, представленная в SEQ ID). NO: 1345) или промотор MND (последовательность представлена в SEQ ID NO: 1361 или 1347. В некоторых вариантах осуществления трансген не включает регуляторный элемент, например промотор.
[0901] Трансген может быть вставлен в эндогенный ген таким образом, что весь эндогенный ген полностью или частично не экспрессируется. В некоторых вариантах осуществления трансген (например, вместе или без пептид-кодирующих последовательностей) интегрируется в любой эндогенный локус. В некоторых вариантах осуществления трансген интегрируется в локусы генов TRAC, TRBC1 и/или TRBC2.
[0902] Кроме того, могут быть включены акцепторные последовательности сплайсинга. Иллюстративные известные последовательности акцепторного сайта сплайсинга включают, например, CTGACCTCTTCTCTTCCTCCCACAG (SEQ ID NO: 1357) (из гена HBB человека) и TTTCTCTCCACAG (SEQ ID NO: 1358) (из гена иммуноглобулина-гамма человека).
[0903] В иллюстративном варианте осуществления матричный полинуклеотид включает в себя плечи гомологии для нацеливания на локус TRAC, регуляторные последовательности, например, промотор, и последовательности нуклеиновых кислот, кодирующие рекомбинантный рецептор, например, TCR. В иллюстративном варианте осуществления используется дополнительный матричный полинуклеотид, который включает в себя плечи гомологии для нацеливания на локусы TRBC1 и/или TRBC2, регуляторные последовательности, например промотор, и последовательности нуклеиновых кислот, кодирующие другой фактор.
[0904] В некоторых вариантах осуществления иллюстративные матричные полинуклеотиды содержат трансген, кодирующий рекомбинантный Т-клеточный рецептор, под функциональным контролем промотора фактора элонгации 1 альфа (EF1α) с энхансером HTLV1 (последовательность представлена в SEQ ID NO: 1345) или промотора MND (последовательность представлена в SEQ ID NO: 1361 или 1347) или связаны с последовательностями нуклеиновых кислот, кодирующими элемент пропуска рибосомы P2A (последовательность представлена в SEQ ID NO: 204), для управления экспрессией рекомбинантного TCR из локуса эндогенного гена-мишени (например, TRAC), 5'-область последовательности плеча гомологии приблизительно 600 п.н. (например, представленная в SEQ ID NO: 1343), 3'-область последовательности плеча гомологии приблизительно 600 п.о. (например, представленная в SEQ ID NO: 1344), которые гомологичны последовательностям, окружающим сайт-мишень интеграции в экзоне 1 гена константной области человеческого TCR α (TRAC). В некоторых вариантах осуществления матричный полинуклеотид дополнительно содержит другие последовательности нуклеиновой кислоты, например последовательности нуклеиновой кислоты, кодирующие маркер, например, поверхностный маркер или маркер селекции. В некоторых вариантах осуществления матричный полинуклеотид дополнительно содержит последовательности вирусного вектора, например, последовательности вектора на основе аденоассоциированного вируса (AAV).
[0905] В некоторых вариантах осуществления трансген дополнительно кодирует один или более маркеров. В некоторых вариантах осуществления один или более маркеров представляют собой маркер трансдукции, суррогатный маркер и/или маркер селекции, включая, но не ограничиваясь этим, любой суррогатный и/или маркер селекции, описанные в настоящем документе.
[0906] В некоторых вариантах осуществления полинуклеотид, например матричный полинуклеотид, содержит последовательность нуклеиновой кислоты, кодирующую фракцию и/или часть рекомбинантного рецептора или его цепи, например рекомбинантного TCR или его цепь, и подвергается направленному воздействию в сайте-мишени (сайтах-мишенях), который находится внутри генного локуса, который кодирует эндогенный рецептор, например, эндогенный ген, кодирующий цепь или домен TCR. В некоторых вариантах осуществления последовательность нуклеиновой кислоты является мишенью для интеграции в рамке внутри локуса эндогенного гена. В конкретных вариантах осуществления интеграция в рамке приводит к получению кодирующей последовательности для рекомбинантного рецептора, которая содержит последовательность нуклеиновой кислоты, кодирующую часть и/или фрагмент рекомбинантного рецептора в рамке с частью и/или фрагментом генного локуса, который кодирует оставшаяся часть и/или фрагмент рецептора, например, для интеграции экзогенной и эндогенной последовательности нуклеиновой кислоты, чтобы получить кодирующую последовательность, кодирующую полный, цельный и/или полноразмерный рекомбинантный рецептор. В определенных вариантах осуществления интеграция генетически нарушает экспрессию эндогенного рецептора, кодируемого геном, в сайте-мишени. В конкретных вариантах осуществления трансген, кодирующий часть рекомбинантного рецептора, направляется в локус гена посредством HDR.
[0907] В некоторых вариантах осуществления трансген кодирует часть рекомбинантного TCR и интегрируется в рамке в эндогенную открытую рамку считывания и/или в локус гена, кодирующего цепь или домен TCR. В некоторых вариантах осуществления трансген кодирует часть рекомбинантного TCR и встраивается в рамке внутрь эндогенной открытой рамки считывания, кодирующей константный домен TCR. В некоторых вариантах осуществления интеграция трансгена в локус модифицирует и/или приводит к получению модифицированного локуса, который кодирует полный рекомбинантный TCR. В конкретных вариантах осуществления часть кодируемого рекомбинантного TCR кодируется последовательностью нуклеиновой кислоты, присутствующей в трансгене, а остальная часть рекомбинантного TCR кодируется последовательностью нуклеиновой кислоты, присутствующей в открытой рамке считывания эндогенного гена, кодирующего константные домены TCRα или TCRβ (например, описанные в Таблицах 13-15 настоящего документа). В конкретных вариантах осуществления транскрипция модифицированного локуса приводит к получению мРНК, которая кодирует рекомбинантный TCR. В конкретных вариантах осуществления часть мРНК транскрибируется из последовательности нуклеиновой кислоты, присутствующей в трансгене, а оставшаяся или дополнительная часть мРНК транскрибируется из последовательности нуклеиновой кислоты, присутствующей в открытой рамке считывания эндогенного гена. В некоторых вариантах осуществления трансген интегрируется в сайт-мишень непосредственно перед и в рамке с областью или частью открытой рамки считывания, которая кодирует оставшуюся часть рекомбинантного TCR.
[0908] В конкретных вариантах осуществления модифицированный локус TRAC или TRBC включает последовательности нуклеиновых кислот, кодирующие рекомбинантный TCR. В некоторых аспектах модифицированный локус TRAC или TRBC в генетически сконструированной клетке содержит трансгенную последовательность (также называемую в настоящем описании как экзогенная или гетерологичная последовательности нуклеиновой кислоты), кодирующую часть рекомбинантного TCR, интегрированного в эндогенный локус TRAC или TRBC, который обычно кодирует константный домен TCRα или TCRβ. В некоторых вариантах осуществления способы включают в себя индукцию направленного генетического нарушения и зависимой от гомологии репарации (HDR) с использованием матричных полинуклеотидов, содержащих трансген, кодирующий часть рекомбинантного TCR, и, таким образом, направляя интеграцию трансгена в локус TRAC или TRBC.
[0909] В некоторых вариантах осуществления трансгенная последовательность, кодирующая часть рекомбинантного TCR, содержит последовательность нуклеотидов, кодирующую цепь TCRβ и часть цепи TCRα. В некоторых вариантах осуществления часть цепи TCRα, кодируемой трансгенными последовательностями, составляет менее чем полноразмерная цепь TCRα. В конкретных вариантах осуществления часть цепи TCRα содержит вариабельный домен TCRα и часть константного домена TCRα, который менее чем полноразмерный константный домен TCRα, например полноразмерный нативный константный домен TCRα, или не содержит последовательности, кодирующей константный домен TCRα. В некоторых аспектах после интеграции трансгенной последовательности в эндогенный локус TRAC полученный модифицированный локус TRAC кодирует рекомбинантный рецептор TCR, кодируемый слиянием трансгена, подвергнутого направленному воздействию с помощью HDR, и открытую рамку считывания или ее частичную последовательность эндогенного локуса TRAC. В некоторых вариантах осуществления кодируемый рекомбинантный TCR содержит цепь TCRα, например, функциональную цепь TCRα, которая способна связываться с цепью TCRβ.
[0910] В конкретных вариантах осуществления трансгенная последовательность, кодирующая часть рекомбинантного TCR, содержит последовательность нуклеотидов, кодирующую цепь TCRα и часть цепи TCRβ. В некоторых вариантах осуществления часть цепи TCRβ, кодируемая трансгенными последовательностями, составляет или включает менее чем полноразмерная цепь TCRβ. В конкретных вариантах осуществления часть цепи TCRβ содержит вариабельный домен TCRβ и часть константного домена TCRβ, который менее чем полноразмерный константный домен TCRβ, например полноразмерный константный домен TCRβ, или не содержит последовательности, кодирующей константный домен TCRβ. В некоторых аспектах после интеграции трансгенной последовательности в эндогенный локус TRBC, например, локус TRBC1 и/или TRBC2, полученный модифицированный локус TRBC кодирует рекомбинантный рецептор TCR, кодируемый слиянием трансгена, нацеленного с помощью HDR, и открытую рамку считывания или ее частичную последовательность эндогенного локуса TRBC. В некоторых вариантах кодированный рекомбинантный TCR содержит цепь TCRβ, например функциональную цепь TCRβ, которая способна связываться с цепью TCRα.
[0911] В конкретных вариантах осуществления рекомбинантный рецептор представляет собой рекомбинантный TCR или его цепь, которая содержит один или более вариабельных доменов и один или более константных доменов. В конкретных вариантах осуществления трансген кодирует часть и/или фрагмент рекомбинантного TCR, который не включает константный домен TCR, и трансген интегрируется в рамке с последовательностью, например последовательностью геномной ДНК, кодирующей константный домен эндогенного TCR. В некоторых вариантах осуществления интеграция приводит к получению кодирующей последовательности, которая кодирует полный, цельный и/или полноразмерный рекомбинантный TCR или его цепь. В некоторых вариантах осуществления кодирующая последовательность содержит трансгенную последовательность, кодирующую часть или фрагмент TCR или его цепь, и эндогенную последовательность, кодирующую константный домен эндогенного TCR.
[0912] В некоторых вариантах осуществления часть рекомбинантного TCR содержит полноразмерную цепь TCRβ (включая вариабельный домен TCRβ и константный домен TCRβ), вариабельный домен TCRα и часть константного домена TCRα. В некоторых вариантах осуществления трансген вставлен или интегрирован в локус TRAC, например, в открытую рамку считывания TRAC, в сайте-мишени, приводя к вставке в рамке трансгена с областью или частью открытой рамки считывания, кодирующей оставшуюся эндогенную часть константного домена TCRα. В некоторых вариантах осуществления вставка приводит к получению модифицированного локуса TRAC, кодирующего полный рекомбинантный TCR. В некоторых вариантах осуществления модифицированный локус TRAC кодирует рекомбинантный TCR, часть которого кодируется последовательностью нуклеиновой кислоты трансгена, такая как часть, которая включает полноразмерную цепь TCRβ (включая вариабельный домен TCRβ и константный домен TCRβ), вариабельный домен TCRα и часть константного домена TCRα и оставшаяся или дополнительная часть, такая как оставшаяся или дополнительная часть константного домена TCRα, кодируется эндогенной и/или нативной последовательностью TRAC. В некоторых вариантах осуществления эндогенная и/или нативная последовательность TRAC кодирует оставшуюся часть рекомбинантного TCR. В некоторых вариантах осуществления матричный полинуклеотид дополнительно содержит другие последовательности нуклеиновой кислоты, например последовательности нуклеиновой кислоты, кодирующие маркер, например, поверхностный маркер или маркер селекции. В некоторых вариантах осуществления матричный полинуклеотид дополнительно содержит последовательности вирусного вектора, например, последовательности вектора на основе аденоассоциированного вируса (AAV).
[0913] В некоторых вариантах осуществления полинуклеотид, например полинуклеотид, такой как матричный полинуклеотид, кодирующий рекомбинантный Т-клеточный рецептор, вводят в клетки в нуклеотидной форме, например, в виде полинуклеотида или вектора. В некоторых вариантах осуществления матричный полинуклеотид вводят в клетку для конструирования, в дополнение к агенту (агентам), способному индуцировать направленное генетическое нарушение, как например нуклеаза и/или гРНК. В некоторых вариантах осуществления матричный полинуклеотид (полинуклеотиды) можно доставлять до, одновременно или после того, как агент (агенты), способный вызывать направленное генетическое нарушение, вводится в клетку. В некоторых вариантах осуществления матричный полинуклеотид (полинуклеотиды) доставляется одновременно с агентами. В некоторых вариантах осуществления матричные полинуклеотиды доставляются перед агентами, например, за секунды и до часов и до дней перед матричными полинуклеотидами, включая, но не ограничиваясь этим, от 1 до 60 минут (или любое время между ними) до агентов, от 1 до 24 часов (или в любое другое время) перед агентами или более чем за 24 часа до агентов. В некоторых вариантах осуществления матричные полинуклеотиды доставляются после агентов, от секунд до часов или до дней после матричных полинуклеотидов, включая момент непосредственно после доставки агента, например, от 1 минуты до 4 часов, например, примерно 10 минут, 20 минут, 30 минут. минуты, 40 минут, 50 минут, 60 минут, 90 минут, 2 часа, 3 часа или 4 часа после доставки агентов и/или предпочтительно в течение 4 часов после доставки агентов. В некоторых вариантах осуществления матричный полинуклеотид доставляется более чем через 4 часа после доставки агентов.
[0914] В некоторых вариантах осуществления матричные полинуклеотиды могут быть доставлены с использованием тех же систем доставки, что и агент (агенты), способный индуцировать направленное генетическое нарушение, например нуклеаза и/или гРНК. В некоторых вариантах осуществления матричные полинуклеотиды могут быть доставлены с использованием систем доставки, отличных от систем доставки агента (агенты), способный индуцировать целевое генетическое нарушение, например нуклеазы и/или гРНК. В некоторых вариантах осуществления матричный полинуклеотид доставляется одновременно с агентом (агентами). В других вариантах осуществления матричный полинуклеотид доставляется в другое время, до или после доставки агента(ов). В некоторых вариантах осуществления любой подходящий способ, известный для введения полинуклеотида в клетку, может быть использован для доставки агентов и/или матричной ДНК, включая описанные в настоящем документе.
[0915] В конкретных вариантах осуществления полинуклеотид, например матричный полинуклеотид, вводится в клетки в нуклеотидной форме, например, в невирусном векторе или внутри него. В некоторых вариантах осуществления невирусный вектор представляет собой или включает полинуклеотид, например полинуклеотид ДНК или РНК, который подходит для трансдукции и/или трансфекции любым подходящим и/или известным невирусным способом доставки генов, таким как, но не ограничиваясь этим, микроинъекция, электропорация, транзиторное сжатие или сдавливание клеток (например, как описано в Lee, et al. (2012) Nano Lett 12: 6322-27), липид-опосредованная трансфекция, пептид-опосредованная доставка, например, проникающие в клетку пептиды или их комбинации.
[0916] В некоторых вариантах осуществления матричная полинуклеотидная последовательность может содержаться в векторной молекуле, содержащей последовательности, которые не гомологичны области, представляющей интерес в геномной ДНК. В некоторых вариантах осуществления вирус представляет собой ДНК-вирус (например, вирус dsДНК или ssДНК). В некоторых вариантах осуществления вирус представляет собой РНК-вирус (например, вирус ssРНК). Иллюстративные вирусные векторы/вирусы включают, например, ретровирусы, лентивирусы, аденовирусы, аденоассоциированные вирусы (AAV), вирусы коровьей оспы, вирусы оспы и вирусы простого герпеса или любые вирусы, описанные где-либо в настоящем документе.
[0917] В некоторых вариантах осуществления матричный полинуклеотид может быть перенесен в клетки с использованием рекомбинантных инфекционных вирусных частиц, таких как, например, векторы, полученные из обезьяньего вируса 40 (SV40), аденовирусов, аденоассоциированного вируса (AAV). В некоторых вариантах осуществления матричный полинуклеотид переносится в Т-клетки с использованием рекомбинантных лентивирусных векторов или ретровирусных векторов, таких как гамма-ретровирусные векторы (см., например, Koste et al. (2014) Gene Therapy 2014 Apr 3. doi: 10.1038/gt.2014.25; Carlens et al. (2000) Exp Hematol 28(10): 1137-46; Alonso-Camino et al. (2013) Mol Ther Nucl Acids 2, e93; Park et al., Trends Biotechnol. 2011 November 29(11): 550-557 или лентивирусные векторы на основе HIV-1.
[0918] В некоторых вариантах осуществления ретровирусный вектор имеет последовательность длинного концевого повтора (LTR), например ретровирусный вектор, полученный из вируса мышиного лейкоза Молони (MoMLV), вируса миелопролиферативной саркомы (MPSV), вируса мышиных эмбриональных стволовых клеток (MESV), мышиного вируса стволовых клеток (MSCV), вируса некроза селезенки (SFFV). Большинство ретровирусных векторов происходят из мышиных ретровирусов. В некоторых вариантах осуществления ретровирусы включают те, которые получены из любого источника клеток птиц или млекопитающих. Ретровирусы, как правило, являются амфотропными, что означает, что они способны инфицировать клетки-хозяева нескольких видов, включая человека. В одном варианте осуществления ген, подлежащий экспрессии, заменяет ретровирусные последовательности gag, pol и/или env. Был описан ряд иллюстративных ретровирусных систем (например, Патенты США No. 5219740; 6207453; 5219740; Miller and Rosman (1989) BioTechniques 7:980-990; Miller, A. D. (1990) Human Gene Therapy 1:5-14; Scarpa et al. (1991) Virology 180:849-852; Burns et al. (1993) Proc. Natl. Acad. Sci. USA 90:8033-8037; и Boris-Lawrie and Temin (1993) Cur. Opin. Genet. Develop. 3: 102-109).
[0919] В других аспектах матричный полинуклеотид доставляется вирусными и/или невирусными методами переноса генов. В некоторых вариантах осуществления матричный полинуклеотид доставляется в клетку через аденоассоциированный вирус (AAV). Может использоваться любой вектор AAV, включая, но не ограничиваясь ими, AAV1, AAV2, AAV3, AAV4, AAV5, AAV6, AAV7, AAV8 и их комбинации. В некоторых случаях AAV содержит LTR, которые имеют гетерологичный серотип по сравнению с серотипом капсида (например, ITR AAV2 с капсидами AAV5, AAV6 или AAV8). Матричный полинуклеотид может быть доставлен с использованием той же системы переноса генов, которая используется для доставки нуклеазы (включая тот же вектор), или может быть доставлен с использованием системы доставки, отличной от той, которая используется для нуклеазы. В некоторых вариантах осуществления матричный полинуклеотид доставляют с использованием вирусного вектора (например, AAV), а нуклеазу (нуклеазы) доставляют в форме мРНК. Клетка также может быть обработана одной или более молекулами, которые ингибируют связывание вирусного вектора с рецептором клеточной поверхности, как описано в настоящем документе, до, одновременно и/или после доставки вирусного вектора (например, несущего нуклеазы (нуклеаз) и/или матричный полинуклеотид).
[0920] В некоторых вариантах осуществления один или более агентов и матричный полинуклеотид доставляются в одном и том же формате или способе. Например, в некоторых вариантах осуществления один или более агентов и матричный полинуклеотид, оба содержатся в векторе, например в вирусном векторе. В некоторых вариантах осуществления матричный полинуклеотид кодируется в одном и том же векторном остове, например геном AAV, плазмидная ДНК, а также Cas9 и гРНК. В некоторых аспектах один или более агентов и матричный полинуклеотид находятся в разных форматах, например, комплекс рибонуклеиновая кислота-белок (RNP) для агента Cas9-гРНК и линейная ДНК для матричного полинуклеотида, но они доставляются одним и тем же методом. В некоторых вариантах осуществления матричные полинуклеотиды и нуклеазы могут находиться в одном и том же векторе, например в векторе AAV (например, AAV6). В некоторых вариантах осуществления матричные полинуклеотиды доставляются с использованием вектора AAV, и агент (агенты), способный индуцировать направленное генетическое нарушение, например, нуклеаза и/или гРНК, доставляются в другой форме, например, в виде мРНК, кодирующих нуклеазы и/или или гРНК. В некоторых вариантах осуществления матричные полинуклеотиды и нуклеазы доставляются с использованием метода того же типа, например, вирусного вектора, но на отдельных векторах. В некоторых вариантах осуществления матричные полинуклеотиды доставляются в другой системе доставки в качестве агентов, способных индуцировать генетическое нарушение, таких как нуклеазы и/или гРНК. В некоторых вариантах осуществления матричный полинуклеотид вырезается из остова вектора in vivo, например, он фланкирован последовательностями распознавания гРНК. В некоторых вариантах осуществления матричный полинуклеотид находится на отдельной полинуклеотидной молекуле, отличной от Cas9 и гРНК. В некоторых вариантах осуществления Cas9 и гРНК вводятся в форме комплекса рибонуклеопротеина (RNP), а матричный полинуклеотид вводится в виде молекулы полинуклеотида, например, в виде вектора или линейной молекулы нуклеиновой кислоты, например, линейной ДНК. Типы или нуклеиновые кислоты и векторы для доставки включают любые из описанных в настоящем документе.
VI. КОМПОЗИЦИИ, СПОСОБЫ И ПРИМЕНЕНИЯ
[0921] Также предложены композиции, включающие связывающие молекулы, например, TCR и сконструированные клетки, включая фармацевтические композиции и составы, и способы применения и применения молекул и композиций, как например при лечении заболеваний, состояний и расстройств, при которых экспрессируется E6 HPV16 или E7 HPV16, и/или детектирование, диагностические и прогностические методы.
A. Фармацевтические композиции и составы
[0922] Предложены фармацевтические составы, включающие связывающие молекулы, например TCR или его антигенсвязывающий фрагмент или антитело или его антигенсвязывающий фрагмент, и/или сконструированные клетки, экспрессирующие связывающие молекулы. Фармацевтические композиции и составы обычно включают один или более необязательных фармацевтически приемлемых носителей или вспомогательных веществ. В некоторых вариантах композиция включает, по меньшей мере, один дополнительный терапевтический агент.
[0923] Термин «фармацевтическая композиция» относится к препарату, который находится в такой форме, которая позволяет биологической активности активного ингредиента, содержащегося в нем, быть эффективной, и который не содержит дополнительных компонентов, которые являются неприемлемо токсичными для пациента, которому композиция будет вводиться.
[0924] «Фармацевтически приемлемый носитель» относится к ингредиенту в фармацевтической композиции, отличному от активного ингредиента, который нетоксичен для пациента. Фармацевтически приемлемый носитель включает, но не ограничивается ими, буфер, вспомогательное вещество, стабилизатор или консервант.
[0925] В некоторых аспектах выбор носителя частично определяется конкретной клеткой или связывающей молекулой и/или способом введения. Соответственно, существует множество подходящих составов. Например, фармацевтическая композиция может содержать консерванты. Подходящие консерванты могут включать, например, метилпарабен, пропилпарабен, бензоат натрия и хлорид бензалкония. В некоторых аспектах используется смесь двух или более консервантов. Консервант или его смеси обычно присутствуют в количестве от примерно 0,0001% до примерно 2% от общей массы композиции. Носители описаны, например, в Remington's Pharmaceutical Sciences 16th edition, Osol, A. Ed. (1980). Фармацевтически приемлемые носители, как правило, нетоксичны для реципиентов в используемых дозировках и концентрациях и включают, но не ограничиваются ими: буферы, такие как фосфат, цитрат и другие органические кислоты; антиоксиданты, включая аскорбиновую кислоту и метионин; консерванты (такие как хлорид октадецилдиметилбензиламмония; хлорид гексаметония; хлорид бензалкония; хлорид бензетония; фенол, бутиловый или бензиловый спирт; алкилпарабены, такие как метил или пропилпарабен; катехол; резорцинол; циклогексанол; 3-пентанол; и м-крезол); низкомолекулярные (менее чем примерно 10 остатков) полипептиды; белки, такие как сывороточный альбумин, желатин или иммуноглобулины; гидрофильные полимеры, такие как поливинилпирролидон; аминокислоты, такие как глицин, глутамин, аспарагин, гистидин, аргинин или лизин; моносахариды, дисахариды и другие углеводы, включая глюкозу, маннозу или декстрины; хелатообразующие агенты, такие как ЭДТА; сахара, такие как сахароза, маннит, трегалоза или сорбит; солеобразующие противоионы, такие как натрий; комплексы металлов (например, комплексы Zn-белок); и/или неионные поверхностно-активные вещества, такие как полиэтиленгликоль (ПЭГ).
[0926] Буферные агенты в некоторых аспектах включены в композиции. Подходящие буферные агенты включают, например, лимонную кислоту, цитрат натрия, фосфорную кислоту, фосфат калия и различные другие кислоты и соли. В некоторых аспектах используется смесь двух или более буферных агентов. Буферный агент или их смеси обычно присутствуют в количестве от примерно 0,001% до примерно 4% от общей массы композиции. Способы приготовления вводимых фармацевтических композиций известны. Примеры способов описаны более подробно, например, в Remington: The Science and Practice of Pharmacy, Lippincott Williams & Wilkins; 21st ed. (May 1, 2005).
[0927] Составы связывающих молекул могут включать лиофилизированные составы и водные растворы. Состав или композиция могут также содержать более чем один активный ингредиент, полезный для конкретного показания, заболевания или состояния, которое лечат связывающими молекулами или клетками, предпочтительно такими, которые обладают активностями, комплементарными связывающей молекуле или клетке, где соответствующие активности не оказывают неблагоприятного воздействия друг на друга. Такие активные ингредиенты подходящим образом присутствуют в комбинации в количествах, которые эффективны для назначенной цели. Таким образом, в некоторых вариантах осуществления фармацевтическая композиция дополнительно включает другие фармацевтически активные агенты или лекарственные средства, такие как химиотерапевтические агенты, например, аспарагиназу, бусульфан, карбоплатин, цисплатин, даунорубицин, доксорубицин, фторурацил, гемцитабин, гидроксимочевину, метотрексат, паклитаксел, ритуксимаб, винбластин, винкристин и т. д. В некоторых вариантах осуществления клетки или связывающие молекулы вводят в форме соли, например, фармацевтически приемлемой соли. Подходящие фармацевтически приемлемые соли присоединения кислоты включают соли, полученные из минеральных кислот, таких как соляная, бромистоводородная, фосфорная, метафосфорная, азотная и серная кислоты, и органических кислот, таких как винная, уксусная, лимонная, яблочная, молочная, фумаровая, бензойная, гликолевая, глюконовая, янтарная и арилсульфоновая кислоты, например п-толуолсульфоновая кислота.
[0928] Активные ингредиенты могут быть заключены в микрокапсулы, в коллоидные системы доставки лекарственных средств (например, липосомы, альбуминовые микросферы, микроэмульсии, наночастицы и нанокапсулы) или в макроэмульсии. В некоторых вариантах осуществления фармацевтическая композиция приготовлена в виде комплекса включения, такого как комплекс включения циклодекстрина, или в виде липосомы. Липосомы могут служить для нацеливания клеток-хозяев (например, Т-клеток или NK-клеток) на конкретную ткань. Для получения липосом доступно много способов, таких как описанные, например, в Szoka et al., Ann. Rev. Biophys. Bioeng., 9: 467 (1980) и в Патентах США 4235871, 4501728, 4837028 и 5019369.
[0929] Фармацевтическая композиция в некоторых аспектах может использовать системы доставки с медленным высвобождением, с отсроченным высвобождением и с замедленным высвобождением, так что доставка композиции происходит до того как и с достаточным временем, чтобы вызвать сенсибилизацию участка, подлежащего лечению. Многие типы систем доставки с высвобождением доступны и известны. Такие системы могут избежать повторного введения композиции, тем самым увеличивая удобство для пациента и врача.
[0930] Фармацевтическая композиция в некоторых вариантах осуществления содержит связывающие молекулы и/или клетки в количествах, эффективных для лечения или предотвращения заболевания или состояния, таких как терапевтически эффективное или профилактически эффективное количество. Терапевтическая или профилактическая эффективность в некоторых вариантах осуществления контролируется путем периодической оценки подвергнутых лечению пациентов. Для повторных введений в течение нескольких дней или дольше, в зависимости от состояния, лечение повторяют до тех пор, пока не произойдет желаемое подавление симптомов заболевания. Тем не менее, другие схемы дозировки могут быть полезны и могут быть определены. Желаемая доза может быть введена однократным болюсным введением композиции, многократным болюсным введением композиции или непрерывным инфузионным введением композиции.
[0931] В некоторых вариантах осуществления, в контексте сконструированных клеток, содержащих связывающие молекулы, пациенту вводят клетки в диапазоне от примерно одного миллиона до примерно 100 миллиардов клеток, например, от 1 миллиона до примерно 50 миллиардов клеток (например, примерно 5 миллионов клеток, примерно 25 миллионов клеток, примерно 500 миллионов клеток, примерно 1 миллиарда клеток, примерно 5 миллиардов клеток, примерно 20 миллиардов клеток, примерно 30 миллиардов клеток, примерно 40 миллиардов клеток или в диапазоне, определенном любыми двумя из вышеуказанных значений), как например от примерно 10 миллионов до примерно 100 миллиардов клеток (например, примерно 20 миллионов клеток, примерно 30 миллионов клеток, примерно 40 миллионов клеток, примерно 60 миллионов клеток, примерно 70 миллионов клеток, примерно 80 миллионов клеток, примерно 90 миллионов клеток, примерно 10 миллиардов клеток, примерно 25 миллиардов клеток, примерно 50 миллиардов клеток, примерно 75 миллиардов клеток, примерно 90 миллиардов клеток или диапазон, определенный любыми двумя из вышеуказанных значений), а в некоторых случаях от примерно 100 миллионов клеток до примерно 50 миллиардов клеток (например, примерно 120 миллионов клеток 250 миллионов клеток, примерно 350 миллионов клеток, примерно 450 миллионов клеток, примерно 650 миллионов клеток, примерно 800 миллионов клеток, примерно 900 миллионов клеток, примерно 3 миллиардов клеток, примерно 30 миллиардов клеток, примерно 45 миллиардов клеток) или любое значение между этими диапазонами и/или такое количество клеток на килограмм массы тела пациента.
[0932] Клетки или связывающие молекулы могут быть введены с использованием стандартных методов введения, составов и/или устройств. Предложены составы и устройства, такие как шприцы и флаконы, для хранения и введения композиций. Введение клеток может быть аутологичным или гетерологичным. Например, иммунореактивные клетки или предшественники могут быть получены от одного пациента и введены одному и тому же пациенту или другому совместимому пациенту. Иммунореактивные клетки периферической крови или их потомство (например, in vivo, ex vivo или in vitro полученные) можно вводить посредством локализованной инъекции, включая введение катетера, системную инъекцию, локализованную инъекцию, внутривенную инъекцию или парентеральное введение. Когда вводят терапевтическую композицию (например, фармацевтическую композицию, содержащую генетически модифицированную иммунореактивную клетку), она, как правило, будет приготовлена в единогй лекарственной форме инъекции (раствор, суспензия, эмульсия).
[0933] Составы включают препараты для перорального, внутривенного, внутрибрюшинного, подкожного, легочного, трансдермального, внутримышечного, интраназального, буккального, сублингвального или суппозиторного введения. В некоторых вариантах осуществления клеточные популяции вводят парентерально. Используемый в настоящем описании термин «парентеральный» включает внутривенное, внутримышечное, подкожное, ректальное, вагинальное, внутричерепное, внутригрудное и внутрибрюшинное введение. В некоторых вариантах осуществления клеточные популяции вводят пациенту с использованием периферической системной доставки путем внутривенной, внутрибрюшинной или подкожной инъекции.
[0934] Композиции в некоторых вариантах осуществления представлены в виде стерильных жидких препаратов, например изотонических водных растворов, суспензий, эмульсий, дисперсий или вязких композиций, которые в некоторых аспектах могут быть забуферены до выбранного рН. Жидкие препараты обычно легче приготовить, чем гели, другие вязкие композиции и твердые композиции. Кроме того, жидкие композиции несколько удобнее вводить, особенно инъекцией. Вязкие композиции, с другой стороны, могут быть приготовлены в пределах соответствующего диапазона вязкости, чтобы обеспечить более длительные периоды контакта с конкретными тканями. Жидкие или вязкие композиции могут содержать носители, которые могут быть растворителем или диспергирующей средой, содержащей, например, воду, физиологический раствор, солевой раствор с фосфатным буфером, полиол (например, глицерин, пропиленгликоль, жидкий полиэтиленгликоль) и их подходящие смеси.
[0935] Стерильные растворы для инъекций могут быть получены путем включения связывающей молекулы в растворитель, как например путем смешивания с подходящим носителем, разбавителем или наполнителем, таким как стерильная вода, физиологический раствор, глюкоза, декстроза или тому подобное. Композиции также могут быть лиофилизированы. Композиции могут содержать вспомогательные вещества, такие как смачивающие, диспергирующие или эмульгирующие агенты (например, метилцеллюлозу), рН-буферные агенты, гелеобразующие или повышающие вязкость добавки, консерванты, ароматизаторы, красители и тому подобное, в зависимости от желаемого пути введения и препарата. В некоторых аспектах можно консультироваться со стандартными текстами, чтобы приготовить подходящие препараты.
[0936] Могут быть добавлены различные добавки, которые повышают стабильность и стерильность композиций, включая антимикробные консерванты, антиоксиданты, хелатообразующие агенты и буферы. Предотвращение действия микроорганизмов может быть обеспечено различными антибактериальными и противогрибковыми средствами, например, парабенами, хлорбутанолом, фенолом, сорбиновой кислотой и тому подобным. Пролонгированная абсорбция инъецируемой фармацевтической формы может быть достигнута путем использования агентов, замедляющих абсорбцию, например моностеарата алюминия и желатина.
[0937] Могут быть приготовлены препараты с замедленным высвобождением. Подходящие примеры препаратов с замедленным высвобождением включают полупроницаемые матрицы из твердых гидрофобных полимеров, содержащих антитело, причем эти матрицы имеют форму профильных изделий, например, пленки или микрокапсулы.
[0938] Составы, используемые для введения in vivo, обычно являются стерильными. Стерильность может быть легко достигнута, например, фильтрацией через стерильные фильтрующие мембраны.
B. Терапевтические и профилактические методы и применения
[0939] Также предложены способы введения и применения, такие как терапевтическое и профилактическое применение связывающих молекул, включая TCR и их антигенсвязывающие фрагменты и антитела или их антигенсвязывающие фрагменты, и/или сконструированных клеток, экспрессирующих связывающие молекулы. Такие способы и применения включают терапевтические способы и применения, например, включающие введение молекул, клеток или композиций, содержащих их, пациенту, имеющему заболевание, состояние или расстройство, экспрессирующее или ассоциированное с HPV, например, HPV16, и/или в которых клетки или ткани экспрессируют, например, специфически экспрессируют, HPV16, например, E6 HPV16 или E7 HPV16. В некоторых вариантах осуществления молекулу, клетку и/или композицию вводят в эффективном количестве для эффективного лечения заболевания или расстройства. Применения включают применения связывающих молекул и клеток в таких способах и лечениях, а также в приготовлении лекарственного средства для осуществления таких терапевтических способов. В некоторых вариантах осуществления способы осуществляются путем введения связывающих молекул или клеток или композиций, содержащих их, пациенту, имеющему, имевшему или у которого подозрение на наличие заболевания или состояния. В некоторых вариантах осуществления способы, таким образом, подвергают лечению заболевание или состояние или расстройство у пациента.
[0940] Используемый в настоящем описании термин «лечение» (и его грамматические вариации, такие как «лечить» или «лечащий») относится к полному или частичному ослаблению или уменьшению заболевания или состояния или расстройства, или симптома, неблагоприятного эффекта или исхода, или связанного с этим фенотипа. Желательные эффекты лечения включают, но не ограничиваются ими, предотвращение возникновения или рецидива заболевания, ослабление симптомов, уменьшение любых прямых или косвенных патологических последствий заболевания, предотвращение метастазирования, уменьшение скорости прогрессирования заболевания, улучшение или смягчение болезненного состояния и ремиссию или улучшение прогноза. Термины не подразумевают полное излечение заболевания или полное устранение какого-либо симптома или воздействия на все симптомы или исходы.
[0941] Используемый в настоящем описании термин «задержка развития заболевания» означает задержать, препятствовать, замедлять, затормозить, стабилизировать, подавить и/или отсрочить развитие заболевания (такого как онкологическое заболевание). Эта задержка может быть различной продолжительности, в зависимости от истории болезни и/или индивидуума, которого подвергают лечению. Как очевидно для специалиста в данной области, достаточная или значительная задержка может, по сути, охватывать предотвращение, поскольку у индивидуума не развивается заболевание. Например, поздняя стадия онкологического заболевания, такая как развитие метастазирования, может быть отсрочена.
[0942] «Предотвращение» при использовании в настоящем описании, включает обеспечение профилактики в отношении возникновения или рецидива заболевания у пациента, который может быть предрасположен к заболеванию, но еще не был диагностирован как имеющий заболевание. В некоторых вариантах осуществления предложенные молекулы и композиции используются для задержки развития заболевания или для замедления прогрессирования заболевания.
[0943] При использовании в настоящем описании, «подавлять» функцию или активность означает уменьшать функцию или активность по сравнению с другими такими же условиями, за исключением состояния или параметра, представляющего интерес, или, альтернативно, по сравнению с другим условием. Например, связывающая молекула или композиция или клетка, которая подавляет рост опухоли, снижает скорость роста опухоли по сравнению со скоростью роста опухоли в отсутствие связывающей молекулы, композиции или клетки.
[0944] «Эффективное количество» агента, например, фармацевтической композиции, связывающей молекулы или клеток или композиции, в контексте введения относится к количеству, эффективному в дозировках/количествах и в течение периодов времени, необходимых для достижения желаемого результата, такого как терапевтический или профилактический результат.
[0945] «Терапевтически эффективное количество» агента, например, фармацевтической композиции, связывающей молекулы или клеток, относится к количеству, эффективному в дозировках и в течение периодов времени, необходимых для достижения желаемого терапевтического результата, как например для лечения заболевания, состояния или расстройства, и/или фармакокинетического или фармакодинамического эффекта лечения. Терапевтически эффективное количество может варьироваться в зависимости от таких факторов, как болезненное состояние, возраст, пол и масса пациента и популяции вводимых клеток. В некоторых вариантах осуществления предложенные способы включают введение связывающих молекул, клеток и/или композиций в эффективных количествах, например терапевтически эффективных количествах.
[0946] «Профилактически эффективное количество» относится к количеству, эффективному в дозировках и в течение периодов времени, необходимых для достижения желаемого профилактического результата. Как правило, но не обязательно, поскольку профилактическая доза используется у пациентов до или на более ранней стадии заболевания, профилактически эффективное количество будет меньше терапевтически эффективного количества.
[0947] Используемый в настоящем описании термин «пациент» представляет собой млекопитающее, такое как человек или другое животное, и обычно представляет собой человека.
[0948] К числу заболеваний, подлежащих лечению, относятся онкологические заболевания, обычно онкологические заболевания, ассоциированные с HPV, и любые ассоциированные с HPV, например, HPV 16-ассоциированные заболевания или состояния, или заболевания или состояния, при которых экспрессируется онкопротеин HPV, например, E6 или E7, такой как онкопротеин HPV 16, например, E6 HPV 16 или E7 HPV 16. При определенных заболеваниях и состояниях вирусный белок, такой как онкопротеин, такой как HPV 16, E6 или E7, экспрессируется в злокачественных клетках или злокачественных новообразованиях или эксрпессируется ими, и/или их пептидный эпитоп экспрессируется на таких злокачественных опухолях или тканях, как например при MHC презентировании. В некоторых вариантах осуществления заболевание или состояние представляет собой злокачественное новообразование, экспрессирующее HPV16. В некоторых вариантах осуществления злокачественное новообразование представляет собой карциному, меланому или другое предраковое или раковое состояние, вызванное или иным образом ассоциированное с HPV, таким как HPV-16. В некоторых вариантах осуществления карцинома может быть плоскоклеточной или аденокарциномой. В некоторых вариантах осуществления заболевание или состояние могут характеризоваться аномалией эпителиальных клеток, связанной с онкогенной инфекцией HPV, такой как килоцитоз; гиперкератоз; предраковые состояния, включающие интраэпителиальные неоплазии или интраэпителиальные поражения; выраженные дисплазии; и инвазивные или злокачественные опухоли. К числу заболеваний или состояний, ассоциированных с HPV 16, которые можно подвергать лечению, относятся, но не ограничиваются ими, рак шейки матки, рак матки, рак анального канала, рак прямой кишки, рак влагалища, рак вульвы, рак полового члена, рак ротоглотки, рак миндалин, фарингеальный рак (рак глотки), рак гортани, рак ротовой полости, рак кожи, рак пищевода, рак головы и шеи, такой как плоскоклеточная карцинома (SCC) или мелкоклеточный рак легких. В некоторых вариантах осуществления заболевание или состояние представляет собой рак шейки матки.
[0949] В некоторых вариантах осуществления способы могут включать в себя стадии или признаки для идентификации пациента, у которого есть подозрение на наличие или риск развития заболевания или расстройства, ассоциированных с HPV 16 (см., например, Патенты США № 6355424 и 8968995), и/или пациент, подлежащий лечению, может быть пациентом, который идентифицирован как имеющий или подверженный такому риску наличия или развития такого HPV-ассоциированного заболевания или состояния или злокачественного новообразования. Следовательно, в некоторых аспектах предложены способы идентификации пациентов с заболеваниями или расстройствами, связанными с экспрессией E6 или E7 HPV 16, и отбора их для лечения и/или лечение таких пациентов, например селективное лечение таких пациентов с помощью предложенной молекулы, связывающей HPV 16, в том числе в некоторых аспектах с клетками, сконструированными для экспрессии таких связывающих молекул, включая в некоторых аспектах любые из TCR E6 или E7 HPV 16 или их антигенсвязывающие фрагменты или антитела против E6 или E7 HPV 16, например фрагменты антител и белки, содержащие их, такие как химерные рецепторы, например TCR-подобные CAR, и/или сконструированные клетки, экспрессирующие TCR или CAR.
[0950] Например, пациент может быть обследован на наличие заболевания или расстройства, ассоциированного с экспрессией E6 или E7 HPV 16, таких как злокачественное новообразование, экспрессирующие E6 или E7 HPV 16. В некоторых вариантах осуществления способы включают в себя скрининг или детектирование присутствия заболевания, ассоциированного с E6 или E7 HPV 16, например, опухоль Таким образом, в некоторых аспектах образец может быть получен от пациента, у которого есть подозрение на заболевание или расстройство, ассоциированное с экспрессией E6 или E7 HPV 16, и проанализирован на уровень экспрессии E6 или E7 HPV 16. В некоторых аспектах пациент, который дает положительный результат на заболевание или расстройство, ассоциированное с E6 или E7 HPV 16, может быть выбран для лечения настоящими способами, и ему может быть введено терапевтически эффективное количество связывающей молекулы, описанной в настоящем документе, CAR, экспрессирующий такую связывающую молекулу, клетки, содержащие связывающую молекулу, или их фармацевтическая композиция, как описано в настоящем документе. В некоторых вариантах осуществления способы могут быть использованы для контроля размера или плотности ткани, экспрессирующей E6 или E7 HPV 16, например, опухоли, со временем, например, до, во время или после лечения способами. В некоторых аспектах пациентов, которых лечат способами, представленными в настоящем документе, выбирают или тестируют как положительных на экспрессию HPV согласно таким способам, например, до начала или во время лечения.
[0951] В некоторых вариантах осуществления введение представленной молекулы, связывающей HPV 16, включая любой из TCR, E6 или E7 HPV 16, или их антигенсвязывающих фрагментов, или антител против E6 или E7 HPV 16, например фрагментов антител и белков, содержащих их, таких как химерные рецепторы, например TCR-подобные CAR, и/или сконструированные клетки, экспрессирующие TCR или CAR, можно комбинировать с другим терапевтическим средством для лечения заболевания HPV. Например, дополнительное терапевтическое лечение может включать лечение другим противоопухолевым средством для лечения рака шейки матки. Подходящие дозировки для такого совместно вводимого агента могут быть снижены благодаря комбинированному действию (синергии) агента и молекулы, связывающей HPV 16.
[0952] В некоторых вариантах осуществления у пациента имеется постоянное или рецидивирующее заболевание, например, после лечения другой молекулой, специфичной для HPV 16, и/или клетками, экспрессирующими нацеленную на HPV 16 связывающую молекулу, и/или другой терапией, включая химиотерапию, облучение и/или трансплантацию гемопоэтических стволовых клеток (HSCT), например, аллогенная HSCT. В некоторых вариантах осуществления введение эффективно лечит пациента, несмотря на то, что пациент становится резистентным к другой HPV 16-направленной терапии. В некоторых вариантах осуществления у пациента не было рецидива, но определено, что он подвержен риску рецидива, например, высокому риску рецидива, и, таким образом, соединение или композицию вводят профилактически, например, для уменьшения вероятности или предотвращения рецидива.
[0953] В некоторых вариантах осуществления лечение не вызывает у пациента иммунного ответа на терапию и/или не вызывает такого ответа в степени, которая препятствует эффективному лечению заболевания или состояния. В некоторых аспектах степень иммуногенности и/или реакции трансплантат против хозяина меньше, чем при другой, но сопоставимой терапии. Например, в случае адоптивной клеточной терапии с использованием клеток, экспрессирующих TCR или CAR, включая предложенные связывающие молекулы, степень иммуногенности в некоторых вариантах осуществления снижается по сравнению с TCR или CAR, включающими другую связывающую молекулу.
[0954] В некоторых вариантах осуществления способы включают адоптивную клеточную терапию, посредством которой сконструированные клетки, экспрессирующие предложенные связывающие молекулы, вводят пациентам. Такое введение может стимулировать HPV 16-направленную активацию клеток (например, активацию Т-клеток), так что клетки заболевания или расстройства подвергаются направленному воздействию для разрушения.
[0955] Таким образом, предложенные способы и применения включают способы и применения для адоптивной клеточной терапии. В некоторых вариантах осуществления способы включают введение клеток или композиции, содержащей клетки, в клетки или ткани пациента, такого как подверженный риску или подозреваемый на наличие заболевания, состояния или расстройства. В некоторых вариантах осуществления клетки, популяции и композиции вводят пациенту, имеющему конкретное заболевание или состояние, подлежащее лечению, например, посредством адоптивной клеточной терапии, такой как адоптивная Т-клеточная терапия. В некоторых вариантах осуществления клетки или композиции вводят пациенту, такому как пациент, имеющий или подверженный риску заболевания или состояния. В некоторых аспектах с помощью способов, таким образом, лечат, например, ослабляют один или более симптомов заболевания или состояния, как например уменьшают опухолевую нагрузку при злокачественном новообразовании, экспрессирующем E6 или E7 HPV 16.
[0956] Способы введения клеток для адоптивной клеточной терапии известны и могут использоваться в связи с предложенными способами и композициями. Например, способы адоптивной Т-клеточной терапии описаны, например, в публикации заявки на патент США № 2003/0170238, выданный Gruenberg et al.; Патент США № 4690915, выданный Rosenberg; Rosenberg (2011) Nat Rev Clin Oncol. 8 (10): 577-85). Смотри, например, Themeli et al. (2013) Nat Biotechnol. 31(10): 928-933; Tsukahara et al. (2013) Biochem Biophys Res Commun 438(1): 84-9; Davila et al. (2013) PLOS ONE 8 (4): e61338.
[0957] В некоторых вариантах осуществления клеточная терапия, например адоптивная клеточная терапия, например адоптивная Т-клеточная терапия, осуществляется путем аутологичного переноса, при котором клетки выделяют и/или иным образом получают от пациента, который должен получить клеточную терапию, или из образца, полученного от такого пациента. Таким образом, в некоторых аспектах клетки получают от пациента, например пациента, нуждающегося в лечении, и клетки после выделения и обработки вводят тому же пациенту.
[0958] В некоторых вариантах осуществления клеточная терапия, например адоптивная клеточная терапия, например адоптивная Т-клеточная терапия, осуществляется путем аллогенного переноса, при котором клетки выделяют и/или иным образом получают от пациента, отличного от пациента, который должен получить или кто в конечном итоге получает клеточную терапию, например, первый пациент. В таких вариантах осуществления клетки затем вводят другому пациенту, например второму пациенту того же вида. В некоторых вариантах осуществления первый и второй пациенты являются генетически идентичными. В некоторых вариантах осуществления первый и второй пациенты генетически сходны. В некоторых вариантах осуществления второй пациент экспрессирует HLA того же класса или супертипа, что и первый пациент.
[0959] В некоторых вариантах осуществления пациент, которому вводят клетки, клеточные популяции или композиции, является приматом, таким как человек. В некоторых вариантах осуществления примат представляет собой обезьяну или человекообразную обезьяну. Пациентом может быть мужчина или женщина и может быть любого подходящего возраста, включая младенцев, юношей, подростков, взрослых и пожилых людей. В некоторых вариантах осуществления пациентом является млекопитающее, не являющимся приматом, такое как грызун. В некоторых примерах пациент или субъект представляет собой одобренную животную модель для заболевания, адоптивной клеточной терапии и/или для оценки токсических результатов, таких как синдром высвобождения цитокинов (CRS).
[0960] Предложенные связывающие молекулы, такие как TCR и их антигенсвязывающие фрагменты, а также антитела и их антигенсвязывающие фрагменты и клетки, экспрессирующие их, можно вводить любым подходящим способом, например, инъекцией, например, внутривенной или подкожной инъекцией, внутриглазной инъекцией, периокулярной инъекцией, субретинальной инъекцией, интравитреальной инъекцией, транс-септальной инъекцией, субсклеральной инъекцией, внутрихориоидальной инъекцией, внутрикамеральной инъекцией, субконъюнктивальной инъекцией, субтеноновой инъекцией, ретробульбарной инъекцией, перибульбарной инъекцией или задней околосклеральной доставкой. В некоторых вариантах осуществления их вводят парентеральным, внутрилегочным и интраназальным, и, если желательно, для местного лечения, введением внутрь пораженного участка. Парентеральные инфузии включают внутримышечное, внутривенное, внутриартериальное, внутрибрюшинное, внутричерепное, внутригрудное или подкожное введение. Дозирование и введение могут частично зависеть от того, является ли введение кратким или хроническим. Различные схемы дозирования включают, но не ограничиваются, однократное или многократное введение в различные моменты времени, болюсное введение и инмпульсную инфузию.
[0961] Для предотвращения или лечения заболевания подходящая дозировка связывающей молекулы или клетки может зависеть от типа заболевания, подлежащего лечению, типа связывающей молекулы, тяжести и течения заболевания, от того, вводится ли связывающая молекула для профилактики или для терапевтических целей, предшествующей терапии, истории болезни пациента и реакции на связывающую молекулу, и призводится по усмотрение лечащего врача. Композиции, молекулы и клетки в некоторых вариантах осуществления соответственно вводят пациенту за один раз или в течение серии процедур.
[0962] В некоторых вариантах осуществления, в контексте сконструированных клеток, содержащих связывающие молекулы, пациенту вводят клетки в диапазоне от примерно одного миллиона до примерно 100 миллиардов клеток, например, от 1 миллиона до примерно 50 миллиардов клеток (например, примерно 5 миллионов клеток, примерно 25 миллионов клеток, примерно 500 миллионов клеток, примерно 1 миллиарда клеток, примерно 5 миллиардов клеток, примерно 20 миллиардов клеток, примерно 30 миллиардов клеток, примерно 40 миллиардов клеток или в диапазоне, определенном любыми двумя из вышеуказанных значений), как например от примерно 10 миллионов до примерно 100 миллиардов клеток (например, примерно 20 миллионов клеток, примерно 30 миллионов клеток, примерно 40 миллионов клеток, примерно 60 миллионов клеток, примерно 70 миллионов клеток, примерно 80 миллионов клеток, примерно 90 миллионов клеток, примерно 10 миллиардов клеток, примерно 25 миллиардов клеток, примерно 50 миллиардов клеток, примерно 75 миллиардов клеток, примерно 90 миллиардов клеток или диапазон, определенный любыми двумя из вышеуказанных значений), а в некоторых случаях от примерно 100 миллионов клеток до примерно 50 миллиардов клеток (например, примерно 120 миллионов клеток 250 миллионов клеток, примерно 350 миллионов клеток, примерно 450 миллионов клеток, примерно 650 миллионов клеток, примерно 800 миллионов клеток, примерно 900 миллионов клеток, примерно 3 миллиардов клеток, примерно 30 миллиардов клеток, примерно 45 миллиардов клеток) или любое значение между этими диапазонами и/или такое количество клеток на килограмм массы тела пациента. Опять же, дозы могут варьироваться в зависимости от особенностей, специфичных для заболевания или расстройства и/или пациента, и/или других способов лечения.
[0963] В некоторых вариантах осуществления связывающие молекулы или клетки вводят как часть комбинированного лечения, как например, одновременно или последовательно, в любом порядке, с другим терапевтическим вмешательством, таким как другой TCR, антитело или сконструированная клетка или рецептор или агент, такой как цитотоксический или терапевтический агент.
[0964] Клетки или антитела в некоторых вариантах осуществления вводят совместно с одним или более дополнительными терапевтическими агентами или в связи с другим терапевтическим вмешательством, либо одновременно, либо последовательно в любом порядке. В некоторых контекстах клетки вводят совместно с другой терапией, достаточно близкой по времени, так что клеточные популяции усиливают действие одного или более дополнительных терапевтических агентов или наоборот. В некоторых вариантах осуществления клетки или антитела вводят до введения одного или более дополнительных терапевтических агентов. В некоторых вариантах осуществления клетки или антитела вводят после введения одного или более дополнительных терапевтических агентов.
[0965] После введения клеток млекопитающему (например, человеку), биологическую активность популяций сконструированных клеток и/или связывающих молекул в некоторых аспектах измеряют любым из ряда известных способов. Параметры для оценки включают специфическое связывание сконструированной или природной Т-клетки или другой иммунной клетки с антигеном in vivo, например, с помощью визуализации или ex vivo, например, с помощью ИФА или проточной цитометрии. В некоторых вариантах осуществления способность сконструированных клеток разрушать клетки-мишени можно измерить, используя любой подходящий метод, известный в данной области, такой как анализы цитотоксичности, описанные, например, в Kochenderfer et al., J. Immunotherapy, 32 (7): 689-702 (2009) и Herman et al. J. Immunological Methods, 285 (1): 25-40 (2004). В некоторых вариантах осуществления биологическая активность клеток также может быть измерена путем анализа экспрессии и/или секреции некоторых цитокинов, таких как CD 107a, IFNγ, IL-2 и TNF. В некоторых аспектах биологическая активность измеряется путем оценки клинического исхода, такого как уменьшение опухолевой массы или нагрузки.
[0966] В некоторых вариантах осуществления сконструированные клетки модифицируют любым количеством способов, так что их терапевтическая или профилактическая эффективность увеличивается. Например, сконструированные TCR или экспрессирующие антитела CAR, экспрессируемые сконструированными клетками в некоторых вариантах осуществления, конъюгированы либо прямо, либо косвенно через линкер с нацеливающим фрагментом. Практика конъюгации соединений, например TCR или CAR, с нацеливающими фрагментами известна в данной области. См., например, Wadwa et al., J. Drug Targeting 3: 11 (1995) и Патент США № 5087616.
C. Методы диагностики и детектирования
[0967] Также предложены способы, включающие использование предложенных связывающих молекул, например TCR или их антигенсвязывающих фрагментов, и антител и их антигенсвязывающих фрагментов, для обнаружения HPV 16, например, E6 HPV 16 или E7 HPV 16, например, в диагностических и/или в прогностических методах в сочетании с HPV 16-экспрессирующим заболеванием или состоянием. Способы в некоторых вариантах осуществления включают инкубацию биологического образца со связывающей молекулой и/или введение связывающей молекулы пациенту. В некоторых вариантах осуществления биологический образец включает клетку или ткань, такую как опухолевая ткань или ткань злокчественного новообразования. В некоторой связывающей молекуле с областью или пептидным эпитопом HPV 16, например E6 или E7 HPV 16, и детектирование того, образуется ли комплекс между связывающей молекулой и пептидным эпитопом. Такой способ может быть методом in vitro или in vivo. В одном варианте осуществления молекула, анти-HPV 16-связывающая молекула используется для отбора пациентов, имеющих право на терапию анти-HPV 16-связывающими молекулами или сконструированными клетками, содержащими такие молекулы, например, где HPV 16, например E6 или E7 HPV 16, представляет собой биомаркер для отбора пациентов.
[0968] В некоторых вариантах осуществления образец, такой как клетка, образец ткани, лизат, композиция или другой полученный из нее образец, контактирует со связывающей молекулой и связывается или образуется комплекс между связывающей молекулой и образцом (например, областью или эпитопом HPV 16 в образце) определяется или детектируется. Когда связывание в тестируемом образце демонстрируется или детектируется по сравнению с контрольной клеткой того же типа ткани, это может указывать на наличие ассоциированного заболевания или состояния. В некоторых вариантах осуществления образец взят из тканей человека.
[0969] Можно использовать различные способы, известные в данной области, для определения специфического связывания молекулы с антигеном. Иллюстративные иммуноанализы включают иммуноанализ с флуоресцентной поляризацией (FPIA), иммуноанализ с флуоресценцией (FIA), иммуноферментный анализ (EIA), иммуноферментный анализ с нефелометрическим ингибированием (NIA), твердофазный иммуноферментный анализ (ELISA) и радиоиммуноанализ (RIA). Индикаторная группа или группа меток может быть присоединена к связывающим молекулам пациента, и может быть выбрана так, чтобы удовлетворить потребности различных применений метода, которые часто продиктованы наличием оборудования для анализа и совместимых процедур иммуноанализа. Иллюстративные метки включают радионуклиды (например, 125I, 131I, 35S, 3H или 32P), ферменты (например, щелочная фосфатаза, пероксидаза хрена, люцифераза или β-глактозидаза), флуоресцентные фрагменты или белки (например, флуоресцеин, родамин, фикоэритрин, GFP или BFP) или люминесцентные фрагменты (например, наночастицы QdotTM, поставляемые компанией Quantum Dot Corporation, Пало-Альто, Калифорния). Общие методики, которые должны использоваться при выполнении различных иммуноанализов, отмеченных выше, известны специалистам в данной области.
[0970] В целях диагностики связывающие молекулы могут быть помечены детектируемым фрагментом, включая, но не ограничиваясь этим, радиоизотопы, флуоресцентные метки и различные фермент-субстратные метки, известные в данной области. Способы конъюгации меток со связывающими молекулами, например TCR или антителами, известны в данной области. В некоторых вариантах осуществления связывающие молекулы не должны быть помечены, и их присутствие может быть детектировано с использованием меченого антитела, которое связывается со связывающими молекулами.
[0971] Предложенные связывающие молекулы в некоторых вариантах осуществления можно использовать в любом известном методе анализа, таком как конкурентные анализы связывания, прямой и непрямой сэндвич-анализы и анализы иммунопреципитации. Связывающие молекулы также можно использовать для диагностических анализов in vivo, таких как визуализация in vivo. Как правило, связывающая молекула помечена радионуклидом (таким как 111In, 99Tc, 14C, 131I, 125I или 3H), так что интересующие клетки или ткани могут быть локализованы in vivo после введения пациенту. Связывающая молекула также может быть использована в качестве окрашивающего реагента при патологии, например, с использованием известных методик.
VII. ПРОМЫШЛЕННЫЕ ИЗДЕЛИЯ
[0972] Также предложены промышленные изделия, содержащие предложенные связывающие молекулы, например, TCR, антитела и CAR и/или сконструированные клетки и/или композиции. Промышленные изделия могут включать контейнер и этикетку или вкладыш в упаковку или связанны с ней. Подходящие контейнеры включают, например, бутылки, флаконы, шприцы, пакеты для внутривенного введения и т. д. Контейнеры могут быть изготовлены из различных материалов, таких как стекло или пластик. Контейнер в некоторых вариантах осуществления содержит композицию, которая представлена сама по себе или объединена с другой композицией, эффективной для лечения, предотвращения и/или диагностики состояния. В некоторых вариантах осуществления контейнер имеет стерильный порт доступа. Иллюстративные контейнеры включают в себя пакеты для внутривенного раствора, флаконы, в том числе с пробками, прокалываемыми иглой для инъекции. Этикетка или вкладыш в упаковку могут указывать, что композиция используется для лечения заболевания или состояния, ассоциированных с Е6 или Е7 HPV 16. Промышленное изделие может включать (а) первый контейнер с содержащейся в нем композицией, где композиция включает антитело или сконструированный антигенный рецептор; и (b) второй контейнер с содержащейся в нем композицией, где композиция включает дополнительный агент, такой как цитотоксический или иной терапевтический агент. Промышленное изделие может дополнительно включать вкладыш в упаковку, указывающий, что композиции можно использовать для лечения конкретного состояния. Альтернативно или дополнительно, промышленное изделие может дополнительно включать другой или такой же контейнер, содержащий фармацевтически приемлемый буфер. Кроме того, он может включать другие материалы, такие как другие буферы, разбавители, фильтры, иглы и/или шприцы.
VIII. Определения
[0973] Если не указано иное, то подразумевается, что все термины в области техники, обозначения и другие технические и научные термины или терминология, используемые в настоящем описании, имеют те же значения, которые обычно понимают специалисты в области техники, к которой относится заявленный объект. В некоторых случаях термины с общепринятыми значениями определены в настоящем документе для ясности и/или для удобства ссылки, и включение таких определений в настоящее описание не обязательно должно истолковываться как представляющее существенную разницу по сравнению с тем, что обычно понимается в данной области техники.
[0974] Термины «полипептид» и «белок» используются взаимозаменяемо для обозначения полимера из аминокислотных остатков и не ограничены минимальной длиной. Полипептиды, включая предложенные антитела и цепи антител и другие пептиды, например линкеры, могут включать аминокислотные остатки, включая природные и/или неприродные аминокислотные остатки. Термины также включают модификации полипептида после экспрессии, например, гликозилирование, сиалилирование, ацетилирование, фосфорилирование и тому подобное. В некоторых аспектах полипептиды могут содержать модификации в отношении нативной или природной последовательности при условии, что белок сохраняет желаемую активность. Эти модификации могут быть преднамеренными, например, путем сайт-направленного мутагенеза, или могут быть случайными, например, в результате мутаций хозяев, которые продуцируют белки, или ошибок вследствие амплификации ПЦР.
[0975] «Выделенная» нуклеиновая кислота относится к молекуле нуклеиновой кислоты, которая была отделена от компонента ее естественной среды. Выделенная нуклеиновая кислота включает молекулу нуклеиновой кислоты, содержащуюся в клетках, которые обычно содержат молекулу нуклеиновой кислоты, но молекула нуклеиновой кислоты присутствует внехромосомно или в хромосомном местоположении, которое отличается от его естественного хромосомного расположения.
[0976] «Выделенная нуклеиновая кислота, кодирующая TCR или антитело» относится к одной или более молекулам нуклеиновой кислоты, кодирующим альфа- или бета-цепи TCR (или их фрагменты) или тяжелые и легкие цепи (или их фрагменты) антитела, включая такие молекулы нуклеиновой кислоты в одном векторе или отдельных векторах, и такие молекулы нуклеиновой кислоты присутствуют в одном или более местах в клетке-хозяине.
[0977] Термины «клетка-хозяин», «линия клеток-хозяев» и «культура клеток-хозяев» используются взаимозаменяемо и относятся к клеткам, в которые была введена экзогенная нуклеиновая кислота, включая потомство таких клеток. Клетки-хозяева включают «трансформанты» и «трансформированные клетки», которые включают первичную трансформированную клетку и ее потомство, независимо от количества пассажей. Потомство может не быть полностью идентичным по содержанию нуклеиновой кислоты родительской клетке, но может содержать мутации. Мутантное потомство, которое имеет ту же функцию или биологическую активность, что и клетки, скринированные или отобранные для первоначальной трансформации, включено в настоящее описание.
[0978] Используемый в настоящем описании термин «процент (%) идентичности аминокислотной последовательности» и «процент идентичности» при использовании в отношении аминокислотной последовательности (эталонной полипептидной последовательности) определяется как процент аминокислотных остатков в последовательности-кандидате (например, рассматриваемое антитело или фрагмент), которые идентичны аминокислотным остаткам в последовательности полипептида сравнения, после выравнивания последовательностей и введения пробелов, если необходимо, для достижения максимальной процентной идентичности последовательности и без учета каких-либо консервативных замен как части идентичности последовательности. Выравнивание в целях определения процента идентичности аминокислотной последовательности может быть достигнуто различными способами, которые известны специалисту в данной области, например, с использованием общедоступного компьютерного программного обеспечения, такого как программное обеспечение BLAST, BLAST-2, ALIGN или Megalign (DNASTAR). Специалисты в данной области могут определить соответствующие параметры для выравнивания последовательностей, включая любые алгоритмы, необходимые для достижения максимального выравнивания по всей длине сравниваемых последовательностей.
[0979] Замена аминокислоты может включать замену одной аминокислоты в полипептиде другой аминокислотой. Аминокислотные замены могут быть введены в связывающую молекулу, например, TCR или антитело, представляющее интерес, и продукты, подвергнутые скринингу на требуемую активность, например, сохраненное/улучшенное связывание антигена, пониженную иммуногенность или улучшенную цитолитическую активность.
[0980] Аминокислоты обычно могут быть сгруппированы в соответствии со следующими общими свойствами боковой цепи:
(1) гидрофобные: норлейцин, Met, Ala, Val, Leu, Ile;
(2) нейтральные гидрофильные: Cys, Ser, Thr, Asn, Gln;
(3) кислые: Asp, Glu;
(4) основные: His, Lys, Arg;
(5) остатки, которые влияют на ориентацию цепи: Gly, Pro;
(6) ароматические: Trp, Tyr, Phe.
[0981] В некоторых вариантах осуществления консервативные замены могут включать замену члена одного из этих классов на другого члена того же класса. В некоторых вариантах осуществления неконсервативные аминокислотные замены могут включать замену члена одного из этих классов на члена другого класса.
[0982] Используемый в настоящем описании термин «вектор» относится к молекуле нуклеиновой кислоты, способной размножать другую нуклеиновую кислоту, с которой она связана. Термин включает вектор как самореплицирующуюся структуру нуклеиновой кислоты, а также вектор, встроенный в геном клетки-хозяина, в которую он был введен. Некоторые векторы способны направлять экспрессию нуклеиновых кислот, с которыми они функционально связаны. Такие векторы упоминаются в настоящем описании как «экспрессирующие векторы».
[0983] Термин «вкладыш в упаковку» используется для обозначения инструкций, обычно включаемых в коммерческие упаковки терапевтических продуктов, которые содержат информацию о показаниях, применении, дозировке, введении, комбинированной терапии, противопоказаниях и/или предупреждениях относительно использования таких терапевтических продуктов.
[0984] При использовании в настоящем описании, формы в единственном числе включают ссылку на множественное число, если контекст явно не предписывает иное. Например, «один» означает «по меньшей мере, один» или «один или более». Понятно, что аспекты и варианты, описанные в настоящем документе, включают «состоящий» и/или «состоящий по существу из» аспектов и вариантов.
[0985] На всем протяжении настоящего раскрытия различные аспекты заявленного объекта представлены в формате диапазона. Следует понимать, что описание в формате диапазона дано просто для удобства и краткости и не должно рассматриваться как жесткое ограничение объема заявленного объекта изобретения. Соответственно, следует считать, что описание диапазона конкретно раскрывает все возможные поддиапазоны, а также отдельные числовые значения в этом диапазоне. Например, если предложен диапазон значений, подразумевается, что каждое промежуточное значение между верхним и нижним пределом этого диапазона и любым другим указанным или промежуточным значением в указанном диапазоне входит в заявленный предмет. Верхний и нижний пределы этих меньших диапазонов могут независимо включаться в меньшие диапазоны и также охватываются заявленным объектом с учетом любого специально исключенного предела в указанном диапазоне. Если указанный диапазон включает один или оба из этих пределов, диапазоны, исключающие один или оба из этих включенных пределов, также включаются в заявленный предмет. Это применяется независимо от широты диапазона.
[0986] Используемый в настоящем описании термин «примерно» относится к обычному диапазону ошибок для соответствующего значения, хорошо известного специалисту в данной области техники. Ссылка на «примерное» значение или параметр в данном документе включает (и описывает) варианты осуществления, которые относятся к этому значению или параметру как таковым. Например, описание, относящееся к «примерно X», включает в себя описание «X».
[0987] Используемый в настоящем описании термин «композиция» относится к любой смеси двух или более продуктов, веществ или соединений, включая клетки. Это может быть раствор, суспензия, жидкость, порошок, паста, водная, неводная или любая их комбинация.
[0988] При использовании в настоящем описании, утверждение о том, что клетка или популяция клеток является «положительной» по конкретному маркеру, относится к детектируемому присутствию на или в клетке определенного маркера, обычно поверхностного маркера. При ссылке на поверхностный маркер термин относится к наличию поверхностной экспрессии, детектируемой с помощью проточной цитометрии, например, путем окрашивания антителом, которое специфически связывается с маркером, и путем детектирования указанного антитела, где окрашивание выявляется с помощью проточной цитометрии на уровне, существенно превышающем детектированное окрашивание, проводя ту же процедуру с соответствующим контролем изотипа в других идентичных условиях, и/или на уровне, по существу аналогичном уровню для клетки, о которой известно, что она является положительной для маркера, и/или на уровне, существенно более высоком, чем для клетки, о которой известной как об отрицательной для маркера.
[0989] При использовании в настоящем описании, утверждение о том, что клетка или популяция клеток является «отрицательной» для конкретного маркера, относится к отсутствию существенного детектируемого присутствия на или в клетке конкретного маркера, обычно поверхностного маркера. При ссылке на поверхностный маркер термин относится к отсутствию поверхностной экспрессии, детектируемой с помощью проточной цитометрии, например, путем окрашивания антителом, которое специфически связывается с маркером, и путем детектирования указанного антитела, где окрашивание не детектируют с помощью проточной цитометрии на уровне, существенно превышающем выявленное окрашивание, выполняя ту же самую процедуру с соответствующим контролем изотипа в других идентичных условиях, и/или на уровне, существенно более низком, чем уровень для клетки, о которой известно, что она является положительной для маркера, и/или на уровне, существенно сходным с уровнем в клетке, которая, как известно, является отрицательной для маркера.
[0990] Все публикации, включая патентные документы, научные статьи и базы данных, упомянутые в настоящей заявке, включены в качестве ссылки во всей их полноте для всех целей в той же степени, как если бы каждая отдельная публикация была отдельно включена в качестве ссылки. Если представленное в настоящем описании определение противоречит или иным образом не согласуется с определением, изложенным в патентах, заявках, опубликованных заявках и других публикациях, которые включены в настоящее описание в качестве ссылки, определение, изложенное здесь, имеет преимущественную силу над определением, которое включено здесь ссылкой,
[0991] Заголовки разделов, используемые в настоящем описании, предназначены только для организационных целей и не должны рассматриваться как ограничивающие предмет описания.
Иллюстративные варианты осуществления
[0992] Среди предложенных вариантов осуществления присутствуют:
1. Т-клеточный рецептор (TCR) или его антигенсвязывающий фрагмент, содержащие альфа-цепь, содержащую вариабельную область альфа (Vα), и бета-цепь, содержащую вариабельную область бета (Vβ), где:
область Vα содержит аминокислотную последовательность, представленную в любой из SEQ ID NO: 691, 709, 726, 741, 759, 775, 787, 799, 815, 830, 845, 857, 869, 881, 895, 908, 925, 937, 951, 963, 975, 987, 999 или 1390, или аминокислотную последовательность, которая имеет с ними по меньшей мере 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% идентичности последовательности; и/или
область Vβ содержит аминокислотную последовательность, представленную в любой из SEQ ID NO: 700, 718, 735, 750, 768, 781, 793, 808, 824, 839, 851, 863, 875, 887, 901, 917, 931, 945, 957, 969, 981, 993, 1008 или 1380, или аминокислотную последовательность, которая имеет с ними по меньшей мере 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% идентичности последовательности.
2. TCR или его антигенсвязывающий фрагмент по варианту осуществления 1, отличающиеся тем, что
область Vα содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность AX2X3X4X5X6X7X8X9X10X11X12X13X14 (SEQ ID NO: 1185), где X2 представляет собой A, G, V, Q, M или E; X3 представляет собой S, G, N, A, Y, R или P; X4 представляет собой E, S, A, G, F, N, D, V, P, L, I, M или R; X5 представляет собой R, N, H, T, D, G, S, P, L, Q или F; X6 представляет собой G, H, A, S, T или отсутствует; X7 представляет собой T, S, G или отсутствует; X8 представляет собой G или отсутствует; X9 представляет собой G, N, S или отсутствует; X10 представляет собой T, G, S, D, F, Y, A или N; X11 представляет собой Y, F, Q, R или N; X12 представляет собой K, Q или D; X13 представляет собой Y, L, T, M, F или V; X14 представляет собой I, T, S, R, Y или V;
область Vα содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность X1X2X3X4X5X6X7X8X9X10KX12I (SEQ ID NO: 1186), где X1 представляет собой A или V; Х2 представляет собой А, V или Е; X3 представляет собой S, N, T, R или P; X4 представляет собой E, A, G, F, V, P, I, D или S; X5 представляет собой R, H, T, A P, S, G или F; X6 представляет собой G, H, L, T, S или A, отсутствует; X7 представляет собой S, T или отсутствует; X8 представляет собой G или отсутствует; X9 представляет собой G, T или отсутствует; Х10 представляет собой F, Y или N; X12 представляет собой Y, T или L;
область Vα содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность AX2X3X4X5X6X7X8X9YKYI (SEQ ID NO: 1187), где X2 представляет собой A, V или E; X3 представляет собой S, N или R; X4 представляет собой E, G, V, P, I или D; X5 представляет собой R, T, P, S, G или F; X6 представляет собой G, T, S или отсутствует; X7 представляет собой S или отсутствует; X8 представляет собой G или отсутствует; Х9 представляет собой Т или отсутствует;
область Vα содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность AX2X3X4X5X6X7X8X9X10X11X12X13X14 (SEQ ID NO: 1188), где X2 представляет собой G, V, Q или M; X3 представляет собой G, A, Y, S, N или R; X4 представляет собой S, G, L, I, M или R; Х5 представляет собой N, D, G, S, L, Q или R; X6 представляет собой A, S, G или отсутствует; X7 представляет собой G или отсутствует; X8 представляет собой G или отсутствует; X9 представляет собой G, N, S или отсутствует; X10 представляет собой S, D, Y, A, N или отсутствует; X11 представляет собой Y, Q или R; Х12 представляет собой К или Q; Х13 представляет собой L или V; X14 представляет собой S, T или V;
область Vα содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность AX2X3X4X5X6X7X8X9X10X11X12X13T (SEQ ID NO: 1189), где X2 представляет собой G, V или Q; X3 представляет собой G, Y, S или N; Х4 представляет собой S, L или М; Х5 представляет собой N, G, L или R; X6 представляет собой A, S, G или отсутствует; X7 представляет собой G или отсутствует; X8 представляет собой G или отсутствует; X9 представляет собой G, S или отсутствует; X10 представляет собой S, Y, A, N или отсутствует; X11 представляет собой Y, Q или R; Х12 представляет собой К или Q; Х13 представляет собой L или V;
область Vα содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность AX2X3X4X5X6X7YKLS (SEQ ID NO: 1190), где X2 представляет собой G или V; Х3 представляет собой А или Y; X4 представляет собой G, S или R; Х5 представляет собой D или S; X6 представляет собой N или отсутствует; X7 представляет собой D или отсутствует.
3. TCR или его антигенсвязывающий фрагмент по варианту осуществления 1 или 2, отличающиеся тем, что
область Vβ содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность AX2X3X4X5X6X7X8X9X10X11X12X13X14 (SEQ ID NO: 1200), где X2 представляет собой S, V или I; X3 представляет собой S, N или A; X4 представляет собой R, V, S, L, P, G, I или A; X5 представляет собой F, G, Y, L, V, R, T или S; X6 представляет собой L, G, A, D, R, V или отсутствует; X7 представляет собой G, D, R, S, T или отсутствует; X8 представляет собой S или отсутствует; X9 представляет собой S, H, G, V, T, D, L или отсутствует; X10 представляет собой T, S, A, G, P, N или Y; X11 представляет собой D, Y, E, G или N; Х12 представляет собой Т, Е, G или К; X13 представляет собой Q, Y или L; X14 представляет собой Y, F, T или I;
область Vβ содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность ASSX4X5X6X7X8X9X10X11X12X13X14 (SEQ ID NO: 1201), где X4 представляет собой R, V, S, L, G или A; X5 представляет собой F, G, Y, L, V, T или S; X6 представляет собой A, L, R, D, G или отсутствует; X7 представляет собой G, D, T или отсутствует; X8 представляет собой S или отсутствует; X9 представляет собой S, H, G, T, D, L или отсутствует; X10 представляет собой T, S, A, G, P, N или Y; X11 представляет собой D, Y, E, G или N; Х12 представляет собой Т, Е или G; X13 представляет собой Q, Y или L; X14 представляет собой Y, F или T;
область Vβ содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность ASSX4X5X6X7X8X9X10TQY (SEQ ID NO: 1202), где X4 представляет собой R, L или G; X5 представляет собой F, V, T или Y; X6 представляет собой L или A, отсутствует; X7 представляет собой G или отсутствует; X8 представляет собой S, G или отсутствует; X9 представляет собой T, G, P или S; X10 представляет собой D или E.
4. Т-клеточный рецептор (TCR) или его антигенсвязывающий фрагмент, содержащие альфа-цепь, содержащую вариабельную область альфа (Vα), и бета-цепь, содержащую вариабельную область бета (Vβ), где:
область Vα содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность AX2X3X4X5X6X7X8X9X10X11X12X13X14 (SEQ ID NO: 1185), где X2 представляет собой A, G, V, Q, M или E; X3 представляет собой S, G, N, A, Y, R или P; X4 представляет собой E, S, A, G, F, N, D, V, P, L, I, M или R; X5 представляет собой R, N, H, T, D, G, S, P, L, Q или F; X6 представляет собой G, H, A, S, T или отсутствует; X7 представляет собой T, S, G или отсутствует; X8 представляет собой G или отсутствует; X9 представляет собой G, N, S или отсутствует; X10 представляет собой T, G, S, D, F, Y, A или N; X11 представляет собой Y, F, Q, R или N; X12 представляет собой K, Q или D; X13 представляет собой Y, L, T, M, F или V; X14 представляет собой I, T, S, R, Y или V;
область Vα содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность X1X2X3X4X5X6X7X8X9X10KX12I (SEQ ID NO: 1186), где X1 представляет собой A или V; Х2 представляет собой А, V или Е; X3 представляет собой S, N, T, R или P; X4 представляет собой E, A, G, F, V, P, I, D или S; X5 представляет собой R, H, T, A P, S, G или F; X6 представляет собой G, H, L, T, S или A, отсутствует; X7 представляет собой S, T или отсутствует; X8 представляет собой G или отсутствует; X9 представляет собой G, T или отсутствует; Х10 представляет собой F, Y или N; X12 представляет собой Y, T или L;
область Vα содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность AX2X3X4X5X6X7X8X9YKYI (SEQ ID NO: 1187), где X2 представляет собой A, V или E; X3 представляет собой S, N или R; X4 представляет собой E, G, V, P, I или D; X5 представляет собой R, T, P, S, G или F; X6 представляет собой G, T, S или отсутствует; X7 представляет собой S или отсутствует; X8 представляет собой G или отсутствует; Х9 представляет собой Т или отсутствует;
область Vα содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность AX2X3X4X5X6X7X8X9X10X11X12X13X14 (SEQ ID NO: 1188), где X2 представляет собой G, V, Q или M; X3 представляет собой G, A, Y, S, N или R; X4 представляет собой S, G, L, I, M или R; Х5 представляет собой N, D, G, S, L, Q или R; X6 представляет собой A, S, G или отсутствует; X7 представляет собой G или отсутствует; X8 представляет собой G или отсутствует; X9 представляет собой G, N, S или отсутствует; X10 представляет собой S, D, Y, A, N или отсутствует; X11 представляет собой Y, Q или R; Х12 представляет собой К или Q; Х13 представляет собой L или V; X14 представляет собой S, T или V;
область Vα содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность AX2X3X4X5X6X7X8X9X10X11X12X13T (SEQ ID NO: 1189), где X2 представляет собой G, V или Q; X3 представляет собой G, Y, S или N; Х4 представляет собой S, L или М; Х5 представляет собой N, G, L или R; X6 представляет собой A, S, G или отсутствует; X7 представляет собой G или отсутствует; X8 представляет собой G или отсутствует; X9 представляет собой G, S или отсутствует; X10 представляет собой S, Y, A, N или отсутствует; X11 представляет собой Y, Q или R; Х12 представляет собой К или Q; Х13 представляет собой L или V;
область Vα содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность AX2X3X4X5X6X7YKLS (SEQ ID NO: 1190), где X2 представляет собой G или V; Х3 представляет собой А или Y; X4 представляет собой G, S или R; Х5 представляет собой D или S; X6 представляет собой N или отсутствует; X7 представляет собой D или отсутствует.
5. Т-клеточный рецептор (TCR) или его антигенсвязывающий фрагмент, содержащие альфа-цепь, содержащую вариабельную область альфа (Vα), и бета-цепь, содержащую вариабельную область бета (Vβ), где:
область Vβ содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность AX2X3X4X5X6X7X8X9X10X11X12X13X14 (SEQ ID NO: 1200), где X2 представляет собой S, V или I; X3 представляет собой S, N или A; X4 представляет собой R, V, S, L, P, G, I или A; X5 представляет собой F, G, Y, L, V, R, T или S; X6 представляет собой L, G, A, D, R, V или отсутствует; X7 представляет собой G, D, R, S, T или отсутствует; X8 представляет собой S или отсутствует; X9 представляет собой S, H, G, V, T, D, L или отсутствует; X10 представляет собой T, S, A, G, P, N или Y; X11 представляет собой D, Y, E, G или N; Х12 представляет собой Т, Е, G или К; X13 представляет собой Q, Y или L; X14 представляет собой Y, F, T или I;
область Vβ содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность ASSX4X5X6X7X8X9X10X11X12X13X14 (SEQ ID NO: 1201), где X4 представляет собой R, V, S, L, G или A; X5 представляет собой F, G, Y, L, V, T или S; X6 представляет собой A, L, R, D, G или отсутствует; X7 представляет собой G, D, T или отсутствует; X8 представляет собой S или отсутствует; X9 представляет собой S, H, G, T, D, L или отсутствует; X10 представляет собой T, S, A, G, P, N или Y; X11 представляет собой D, Y, E, G или N; Х12 представляет собой Т, Е или G; X13 представляет собой Q, Y или L; X14 представляет собой Y, F или T;
область Vβ содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность ASSX4X5X6X7X8X9X10TQY (SEQ ID NO: 1202), где X4 представляет собой R, L или G; X5 представляет собой F, V, T или Y; X6 представляет собой L или A, отсутствует; X7 представляет собой G или отсутствует; X8 представляет собой S, G или отсутствует; X9 представляет собой T, G, P или S; X10 представляет собой D или E.
6. Т-клеточный рецептор (TCR) или его антигенсвязывающий фрагмент, содержащие альфа-цепь, содержащую вариабельную область альфа (Vα), и бета-цепь, содержащую вариабельную область бета (Vβ), где:
область Vα содержит область 3, определяющую комплементарность, (CDR-3), представленную в любой из SEQ ID NO: 694, 712, 729, 744, 762, 776, 788, 802, 818, 832, 846, 858, 870, 882, 896, 911, 926, 940, 952, 964, 976, 988, 1002 или последовательности, которая имеет с ними по меньшей мере 60%, 65%, 70%, 75%, 80%, 85%, 90% или 95% идентичности последовательности;
область Vβ содержит область 3, определяющую комплементарность, (CDR-3), представленную в любой из SEQ ID NO: 703, 721, 736, 753, 769, 782, 794, 809, 825, 840, 852, 864, 876, 888, 902, 919, 932, 946, 958, 970, 982, 994 или 1010 или последовательности, которая имеет с ними по меньшей мере 60%, 65%, 70%, 75%, 80%, 85%, 90% или 95% идентичности последовательности.
7. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 1-6, где область Vα содержит:
область 1, определяющую комплементарность, (CDR-1), содержащую аминокислотную последовательность X1X2X3X4X5X6 (SEQ ID NO: 1191), где X1 представляет собой N, S, D, T или V; X2 представляет собой S, V, R, T или I; Х3 представляет собой М, F, G, S, N, А, L, V или Р; X4 представляет собой F, S, N, A или отсутствует; X5 представляет собой D, S, Q, Y, N, V, T или P; и X6 представляет собой Y, S, R, N, G или T; и/или
область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность X1X2X3X4X5X6X7X8 (SEQ ID NO: 1192), где X1 представляет собой I, V, L, G, N, T, Y или M; X2 представляет собой S, V, Y, L, P, F, I или T; X3 представляет собой S, Y, K, L, T или F; X4 представляет собой I, G, N, A, S или отсутствует; X5 представляет собой S, D или отсутствует; X6 представляет собой K, G, N, S, D, T или E; X7 представляет собой D, E, G, A, K, L или N; и X8 представляет собой K, V, D, P, N, T, L или M.
8. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 1-7, где область Vβ содержит:
область 1, определяющую комплементарность, (CDR-1), содержащую аминокислотную последовательность SX2X3X4X5 (SEQ ID NO: 1203), где X2 представляет собой G или N; Х3 представляет собой Н или D; Х4 представляет собой Т, L, N или V; и X5 представляет собой A, S, Y или T; и/или
область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность X1X2X3X4X5X6 (SEQ ID NO: 1204), где X1 представляет собой F или Y; Х2 представляет собой Q, Y или N; X3 представляет собой G, N, R или Y; X4 представляет собой N, G, E или T; X5 представляет собой S, E, A или G; и X6 представляет собой A, E, I или Q.
9. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 1-8, где TCR или его антигенсвязывающий фрагмент связываются или распознают пептидный эпитоп вируса папилломы человека (HPV) 16 E7 в контексте молекулы МНС, причем пептидный эпитоп представляет собой или содержит Е7 (11-19) YMLDLQPET (SEQ ID NO: 236).
10. TCR или антигенсвязывающий фрагмент по любому из вариантов осуществления 1-9, отличающиеся тем, что:
область Vα содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 694, 712, 729, 744, 762, 776, 788, 802, 818, 832, 846, 858, 870, 882, 896, 911, 926, 940, 952, 964, 976, 988 или 1002, или CDR3, содержащуюся в аминокислотной последовательности, представленной в любой из SEQ ID NO: 691, 709, 726, 741, 759, 775, 787, 799, 815, 830, 845, 857, 869, 881, 895, 908, 925, 937, 951, 963, 975, 987 или 999; и/или
область Vβ содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 703, 721, 736, 753, 769, 782, 794, 809, 825, 840, 852, 864, 876, 888, 902, 919, 932, 946, 958, 970, 982, 994, 1010 или 1381, или CDR3, содержащуюся в аминокислотной последовательности, представленной в любой из SEQ ID NO: 700, 718, 735 750, 768, 781, 793, 808, 824, 839, 851, 863, 875, 887, 901, 917, 931, 945, 957, 969, 981, 993, 1008 или 1380.
11. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 1-10, где область Vα дополнительно содержит:
область 1, определяющую комплементарность, (CDR-1), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 171, 692, 710, 727, 742, 760, 171, 800, 816, 570, 909, 938, 151, или 1000; и/или
область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 172, 693, 711, 728, 743, 761, 172, 801, 817, 831, 571, 910, 939, 152 или 1001.
12. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 1-11, где область Vβ содержит:
область 1, определяющую комплементарность, (CDR-1), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 701, 719, 154, 751 или 139; и/или
область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 702, 720, 155, 752, 140 или 918.
13. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 1-12, отличающиеся тем, что
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 692, 693 и 694, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 701, 702 и 703, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 710, 711 и 712, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 719, 720 и 721, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 727, 728 и 729, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 736, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 742, 743 и 744, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 751, 752 и 753, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 760, 761 и 762, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 719, 720 и 769, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 171, 172 и 776, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 782, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 742, 743 и 788, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 139, 140 и 794, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 800, 801 и 802, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 751, 752 и 809, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 816, 817 и 818, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 825, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 816, 831 и 832, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 840, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 171, 172 и 846, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 852, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 816, 831 и 858, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 864, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 727, 728 и 870, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 876, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 570, 571 и 882, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 719, 720 и 888, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 816, 817 и 896, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 701, 702 и 902, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 909, 910 и 911, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 701, 702 и 919, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 727, 728 и 926, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 932, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 938, 939 и 940, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 946, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 727, 728 и 952, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 958, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 151, 152 и 964, соответственно, а область Vβ содержит CDR-1, CDR-2 и CDR -3, содержащие аминокислотные последовательности SEQ ID NO: 719, 720 и 970, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 727, 728 и 976, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 982, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 710, 711 и 988, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 719, 729 и 994, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 1000, 1001 и 1002, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 139, 1009 и 1010, соответственно; или
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 171, 172 и 1391, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 1381, соответственно.
14. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 1-13, отличающиеся тем, что
область Vα содержит область 1, определяющую комплементарность, (CDR-1), CDR-2 и CDR-3, соответственно, содержащие аминокислотные последовательности CDR-1, CDR-2 и CDR-3, содержащиеся в аминокислотной последовательности области Vα, представленной в любой из SEQ ID NO: 691, 709, 726, 741, 759, 775, 787, 799, 815, 830, 845, 857, 869, 881, 895, 908, 925, 937, 951, 963 975, 987, 999 или 1390; и/или
область Vβ содержит область 1, определяющую комплементарность, (CDR-1), CDR-2 и CDR-3, соответственно, содержащие аминокислотные последовательности CDR-1, CDR-2 и CDR-3, содержащиеся в аминокислотной последовательности области Vβ, представленной в любой из SEQ ID NO: 700, 718, 735, 750, 768, 781, 793, 808, 824, 839, 851, 863, 875, 887, 901, 917, 931, 945, 957, 969 981, 993, 1008 или 1380.
15. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 1-14, отличающиеся тем, что
области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 691 и 700, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 709 и 718, соответственно; области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 726 и 735, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 741 и 750, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 759 и 768, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 775 и 781, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 787 и 793, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 799 и 808, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 815 и 824, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 830 и 839, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 845 и 851, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 857 и 863, соответственно, режим Vα и Vβ он включает аминокислотные последовательности SEQ ID NO: 869 и 875, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 881 и 887, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 895 и 901, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 908 и 917, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 925 и 931, соответственно области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 937 и 945, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 951 и 957, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 963 и 969, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 975 и 981, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 987 и 993, соответственно; области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 999 и 1008, соответственно; или области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 1390 и 1380, соответственно.
16. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 1-15, где альфа-цепь дополнительно содержит константную область альфа (Cα), и/или бета-цепь дополнительно содержит константную область бета (Cβ).
17. TCR или его антигенсвязывающий фрагмент по варианту осуществления 16, где области Cα и Cβ представляют собой константные области мыши.
18. TCR или его антигенсвязывающий фрагмент по варианту осуществления 16 или 17, отличающиеся тем, что
область Cα содержит аминокислотную последовательность, представленную в SEQ ID NO: 262, 833, 1012, 1014, 1015, 1017, 1018, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности; и/или
область Cβ содержит аминокислотную последовательность, представленную в SEQ ID NO: 263, 1013 или 1016, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности.
19. TCR или его антигенсвязывающий фрагмент по варианту осуществления 16, где области Cα и Cβ представляют собой константные области человека.
20. TCR или его антигенсвязывающий фрагмент по варианту осуществления 16 или 19, отличающиеся тем, что
область Cα содержит аминокислотную последовательность, представленную в любой из SEQ ID NO: 212, 213, 215, 217, 218, 220 или 524, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности; и/или
область Cβ содержит аминокислотную последовательность, представленную в любой из SEQ ID NO: 214, 216, 631 или 889, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности.
21. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 1-20, отличающиеся тем, что
а) альфа-цепь содержит:
аминокислотную последовательность, представленную в любой из SEQ ID NO: 687, 705, 722, 737, 755, 771, 783, 795, 811, 826, 841, 853, 865, 877, 891, 904, 921, 933, 947 959, 971, 983, 995, 1386, последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности; или аминокислотную последовательность, кодируемую нуклеотидной последовательностью, представленной в любой из SEQ ID NO: 1049, 1051, 1055, 1057, 1059, 1061, 1063, 1065, 1067, 1069, 1071, 1073, 1075, 1075, 1077, 1079, 1081, 1083, 1085, 1087, 1089, 1091, или нуклеотидной последовательностью, которая имеет с ними по меньшей мере 90% идентичности последовательности; и/или
b) бета-цепь содержит:
аминокислотную последовательность, представленную в любой из SEQ ID NO: 696, 714, 731, 746, 764, 777, 789, 804, 820, 835, 847, 859, 871, 883, 897, 913, 913, 927, 941, 953, 965, 977, 989, 1004 или 1376, последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности; или аминокислотную последовательность, кодируемую нуклеотидной последовательностью, представленной в SEQ ID NO: 1050, 1052, 1056, 1058, 1060, 1062, 1064, 1066, 1068, 1070, 1072, 1074, 1076, 1078, 1080, 1082, 1084, 1086, 1088, 1090 или 1092, или нуклеотидной последовательностью, которая имеет с ними по меньшей мере 90% идентичности последовательности.
22. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 1-20, отличающиеся тем, что
альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 687 и 696, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 705 и 714, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 722 и 731, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 737 и 746, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 755 и 764, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 771 и 777, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 783 и 789, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 795 и 804, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 811 и 820, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 826 и 835, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 841 и 847, соответственно, альфа и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 853 и 859, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 865 и 871, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 877 и 883, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 891 и 897, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 904 и 913, соответственно, альфа- и бета-цепи включают аминокислотные последовательности SEQ ID NO: 921 и 927, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 933 и 941, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 947 и 953, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 959 и 965, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 971 и 977, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 983 и 989, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 995 и 1004, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 1386 и 1376, соответственно.
23. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 1-19, где TCR или антигенсвязывающий фрагмент содержат одну или более модификаций в α-цепи и/или β-цепи, так что когда TCR или его антигенсвязывающий фрагмент экспрессируются в клетке, то снижается частота ошибочных спариваний между α-цепью и β-цепью TCR и α-цепью и β-цепью эндогенного TCR, экспрессия α-цепи и β-цепи TCR увеличивается и/или стабильность α цепи и β-цепи TCR увеличивается, каждая по сравнению с экспрессией в клетке TCR или его антигенсвязывающего фрагмента, не содержащих одну или более модификаций.
24. TCR или его антигенсвязывающий фрагмент по варианту осуществления 23, где одна или более модификаций представляют собой замену, делецию или вставку одной или более аминокислот в области Cα; и/или области Cα.
25. TCR или его антигенсвязывающий фрагмент по варианту осуществления 23 или 24, где одна или более модификаций содержат замену(замены) для введения одного или более остатков цистеина, которые способны образовывать одну или более ненативных дисульфидных связей между альфа-цепью и бета-цепью.
26. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 1-16, 19 и 23-25, содержащие область Cα, содержащую цистеин в положении, соответствующем положению 48, с нумерацией, представленной в SEQ ID NO: 212, 213, 217, 218 или 524, или в положении, соответствующем положению 49 с нумерацией, представленной в SEQ ID NO: 215 или 220; и/или область Cβ, содержащую цистеин в положении, соответствующем положению 57 с нумерацией, представленной в SEQ ID NO: 214 или 216, или в положении, соответствующем положению 58 с нумерацией, представленной в SEQ ID NO: 631 или 889.
27. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 16, 19 и 23-26, отличающиеся тем, что:
область Cα содержит аминокислотную последовательность, представленную в любой из SEQ ID NO: 196, 198, 200, 201, 203 или 525, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности, содержащие один или более остатков цистеина, способных образовывать ненативную дисульфидную связь с бета-цепью; и/или
область Cβ содержит аминокислотную последовательность, представленную в любой из SEQ ID NO: 197, 199, 632 или 890, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности, которые содержат один или более остатков цистеина, способных образовывать ненативную дисульфидную связь с альфа-цепью.
28. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 1-27, где TCR или его антигенсвязывающий фрагмент кодируются нуклеотидной последовательностью, которая была оптимизирована по кодонам.
29. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 1-19 и 23-28, отличающиеся тем, что
а) альфа-цепь содержит:
аминокислотную последовательность, представленную в любой из SEQ ID NO: 688, 706, 723, 738, 756, 772, 784, 796, 812, 827, 842, 854, 866, 878, 892, 905, 922, 934, 948, 960, 972, 984, 996 или 1387, последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности; или аминокислотную последовательность, кодируемую нуклеотидной последовательностью, представленной в любой из SEQ ID NO: 1129, 1131, 1133, 1135, 1137, 1139, 1141, 1143, 1145, 1147, 1149, 1151, 1153, 1155, 1157, 1159, 1161, 1163, 1165, 1167, 1169, 1171, 1173 или 1385, или нуклеотидной последовательностью, которая имеет с ними по меньшей мере 90% идентичности последовательности; и/или
b) бета-цепь содержит:
аминокислотную последовательность, представленную в любой из SEQ ID NO: 697, 715, 732, 747, 765, 778, 790, 805, 821, 836, 848, 860, 872, 884, 898, 914, 928, 942, 954, 966, 978, 990, 1005 или 1377, последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности; или аминокислотную последовательность, кодируемую нуклеотидной последовательностью, представленной в SEQ ID NO: 1130, 1132, 1134, 1136, 1138, 1140, 1142, 1144, 1146, 1148, 1150, 1152, 1154, 1156, 1158, 1160, 1162, 1164, 1166, 1168, 1170, 1172, 1174 или 1375, или нуклеотидной последовательностью, которая имеет с ними по меньшей мере 90% идентичности последовательности.
30. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 1-19 и 23-29, отличающиеся тем, что:
альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 688 и 697, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 706 и 715, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 723 и 732, соответственно, альфа- и бета-цепи содержат амино кислотные последовательности SEQ ID NO: 738 и 747, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 756 и 765, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 772 и 778, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 784 и 790, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 796 и 805, соответственно, альфа и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 812 и 821, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 827 и 836, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 842 и 848, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 854 и 860, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 866 и 872, соответственно, альфа- и бета-цепи включают аминокислотные последовательности SEQ ID NO: 878 и 884, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 892 и 898, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 905 и 914, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 922 и 928, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 934 и 942, соответственно, альфа- и бета цепи содержат аминокислотные последовательности SEQ ID NO: 948 и 954, соответственно, альфа- и бета цепи содержат аминокислотные последовательности SEQ ID NO: 960 и 966, соответственно, альфа- и бета цепи содержат аминокислотные последовательности SEQ ID NO: 972 и 978, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 984 и 990, соответственно; или альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 996 и 1005, соответственно; или альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 1387 и 1377, соответственно.
31. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 1-30, где альфа- и/или бета-цепь дополнительно содержат сигнальный пептид.
32. TCR или его антигенсвязывающий фрагмент по варианту осуществления 31, отличающиеся тем, что:
альфа-цепь содержит сигнальный пептид, содержащий аминокислотную последовательность, представленную в любой из SEQ ID NO: 181, 184, 187, 189, 190, 192, 193, 310, 311; и/или
бета-цепь содержит сигнальный пептид, содержащий аминокислотную последовательность, представленную в любой из SEQ ID NO: 182, 185, 186, 188, 191 или 194.
33. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 1-32, которые выделены или очищены или являются рекомбинантными.
34. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 1-33, которые являются человеческими.
35. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 1-34, которые являются моноклональными.
36. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 1-35, где TCR или его антигенсвязывающий фрагмент являются одноцепочечными.
37. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 1-35, где TCR или его антигенсвязывающий фрагмент содержат две цепи.
38. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 1-37, где антигенспецифичность является по меньшей мере частично CD8-независимой.
39. TCR или антигенсвязывающий фрагмент по любому из вариантов осуществления 9-38, где молекула МНС представляет собой молекулу HLA-A2.
40. Молекула нуклеиновой кислоты, кодирующая TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 1-39 или его альфа- или бета-цепь.
41. Молекула нуклеиновой кислоты по варианту осуществления 40, содержащая нуклеотидную последовательность, кодирующую альфа-цепь, и/или нуклеотидную последовательность, кодирующую бета-цепь, отличающаяся тем, что:
нуклеотидная последовательность, кодирующая альфа-цепь, содержит последовательность, представленную в любой из SEQ ID NO: 1049, 1051, 1055, 1057, 1059, 1061, 1063, 1065, 1067, 1069, 1071, 1073, 1075, 1075, 1077, 1079, 1081, 1083, 1085, 1087, 1089, 1091, или нуклеотидную последовательность, которая имеет с ними по меньшей мере 90% идентичности последовательности;
нуклеотидная последовательность, кодирующая бета-цепь, содержит последовательность, представленную в SEQ ID NO: 1050, 1052, 1056, 1058, 1060, 1062, 1064, 1066, 1068, 1070, 1072, 1074, 1076, 1078, 1080, 1082, 1084, 1086, 1088, 1090 или 1092, или нуклеотидную последовательность, которая имеет с ними по меньшей мере 90% идентичности последовательности.
42. Молекула нуклеиновой кислоты по варианту осуществления 40, где нуклеотидная последовательность оптимизирована по кодонам.
43. Молекула нуклеиновой кислоты по варианту осуществления 40 или 42, содержащая нуклеотидную последовательность, кодирующую альфа-цепь, и/или нуклеотидную последовательность, кодирующую бета-цепь, отличающаяся тем, что:
нуклеотидная последовательность, кодирующая альфа-цепь, содержит последовательность, представленную в любой из SEQ ID NO: 1129, 1131, 1133, 1135, 1137, 1139, 1141, 1143, 1145, 1147, 1149, 1151, 1153, 1155, 1157, 1159, 1161, 1163, 1165, 1167, 1169, 1171, 1173 или 1385, или нуклеотидную последовательность, которая имеет с ними по меньшей мере 90% идентичности последовательности;
нуклеотидная последовательность, кодирующая бета-цепь, содержит последовательность, представленную в SEQ ID NO: 1130, 1132, 1134, 1136, 1138, 1140, 1142, 1144, 1146, 1148, 1150, 1152, 1154, 1156, 1158, 1160, 1162, 1164, 1166, 1168, 1170, 1172, 1174 или 1375, или нуклеотидную последовательность, которая имеет с ними по меньшей мере 90% идентичности последовательности.
44. Молекула нуклеиновой кислоты по любому из вариантов осуществления 40-43, где нуклеотидная последовательность, кодирующая альфа-цепь, и нуклеотидная последовательность, кодирующая бета-цепь, разделены пептидной последовательностью, которая вызывает пропуск рибосомы.
45. Молекула нуклеиновой кислоты по варианту осуществления 44, где пептид, который вызывает пропуск рибосомы, представляет собой пептид P2A или T2A и/или содержит последовательность аминокислот, представленную в SEQ ID NO: 204 или 211.
46. Молекула нуклеиновой кислоты по любому из вариантов осуществления 40-45, содержащая нуклеотидную последовательность, представленную в любой из SEQ ID NO: 448, 449, 450, 451, 452, 453, 454, 455, 456, 457, 458, 459, 460. 461, 462, 463, 464, 465, 466, 467, 468, 469, 470, 471, 472 или 1382, или нуклеотидную последовательность, имеющую с ними по меньшей мере 90% идентичности последовательности.
47. Т-клеточный рецептор (TCR) или его антигенсвязывающий фрагмент, содержащие альфа-цепь, содержащую вариабельную область альфа (Vα), и бета-цепь, содержащую вариабельную область бета (Vβ), где:
область Vα содержит аминокислотную последовательность, представленную в любой из SEQ ID NO: 477, 492, 504, 510, 522, 536, 554, 569, 587, 599, 611, 623, 637, 649, 661 или 676, или аминокислотную последовательность, которая имеет с ними по меньшей мере 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% идентичности последовательности; и/или
область Vβ содержит аминокислотную последовательность, представленную в любой из SEQ ID NO: 483, 498, 516, 530, 545, 560, 578, 593, 605, 617, 629, 643, 655, 667 или 685, или аминокислотную последовательность, которая имеет с ними по меньшей мере 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% идентичности последовательности.
48. TCR или его антигенсвязывающий фрагмент по варианту осуществления 47, где область Vα содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность AX2RX4AX6NNDMR, где X2 представляет собой V или M; Х4 представляет собой Р или D; и X6 представляет собой N или R.
49. TCR или его антигенсвязывающий фрагмент по варианту осуществления 47 или 48, отличающиеся тем, что:
область Vβ содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность ASSX4WGX7SNQPX12H, где X4 представляет собой L, F или P; X7 представляет собой R или Q; и X12 представляет собой Q или L; или
область Vβ содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность ASSX4X5X6X7X8SGNTIY, где X4 представляет собой L или R; Х5 представляет собой W или Q; X6 представляет собой G или P; Х7 представляет собой R или S; и X8 представляет собой S или отсутствует.
50. T-клеточный рецептор (TCR) или его антигенсвязывающий фрагмент, содержащие альфа-цепь, содержащую вариабельную область альфа (Vα), и бета-цепь, содержащую вариабельную область бета (Vβ), где область Vα содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность AX2RX4AX6NNDMR, где X2 представляет собой V или М; Х4 представляет собой Р или D; и X6 представляет собой N или R.
51. Т-клеточный рецептор (TCR) или его антигенсвязывающий фрагмент, содержащие альфа-цепь, содержащую вариабельную область альфа (Vα), и бета-цепь, содержащую вариабельную область бета (Vβ), где:
область Vβ содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность ASSX4WGX7SNQPX12H, где X4 представляет собой L, F или P; X7 представляет собой R или Q; и X12 представляет собой Q или L; или
область Vβ содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность ASSX4X5X6X7X8SGNTIY, где X4 представляет собой L или R; Х5 представляет собой W или Q; X6 представляет собой G или P; Х7 представляет собой R или S; и X8 представляет собой S или отсутствует.
52. Т-клеточный рецептор (TCR) или его антигенсвязывающий фрагмент, содержащие альфа-цепь, содержащую вариабельную область альфа (Vα), и бета-цепь, содержащую вариабельную область бета (Vβ), где:
область Vα содержит область 3, определяющую комплементарность, (CDR-3), представленную в любой из SEQ ID NO: 478, 493, 505, 511, 523, 539, 555, 572, 588, 600, 612, 624, 638, 650, 662 или 679, или в последовательности, которая имеет с ними по меньшей мере 60%, 65%, 70%, 75%, 80%, 85%, 90% или 95% идентичности последовательности;
область Vβ содержит область 3, определяющую комплементарность, (CDR-3), представленную в любой из SEQ ID NO: 486, 499, 517, 531, 548, 563, 581, 594, 606, 618, 630, 644, 656, 670 или 686, или в последовательности, которая имеет с ними по меньшей мере 60%, 65%, 70%, 75%, 80%, 85%, 90% или 95% идентичности последовательности.
53. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 47-52, где область Vα содержит:
область 1, определяющую комплементарность, (CDR-1), содержащую аминокислотную последовательность X1X2X3X4X5X6 (SEQ ID NO: 1191), где X1 представляет собой N, S, D, T или V; X2 представляет собой S, V, R, T или I; Х3 представляет собой М, F, G, S, N, А, L, V или Р; X4 представляет собой F, S, N, A или отсутствует; X5 представляет собой D, S, Q, Y, N, V, T или P; и X6 представляет собой Y, S, R, N, G или T; и/или
область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность X1X2X3X4X5X6X7X8 (SEQ ID NO: 1192), где X1 представляет собой I, V, L, G, N, T, Y или M; X2 представляет собой S, V, Y, L, P, F, I или T; X3 представляет собой S, Y, K, L, T или F; X4 представляет собой I, G, N, A, S или отсутствует; X5 представляет собой S, D или отсутствует; X6 представляет собой K, G, N, S, D, T или E; X7 представляет собой D, E, G, A, K, L или N; и X8 представляет собой K, V, D, P, N, T, L или M.
54. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 47-53, где область Vβ содержит:
область 1, определяющую комплементарность, (CDR-1), содержащую аминокислотную последовательность SX2X3X4X5 (SEQ ID NO: 1203), где X2 представляет собой G или N; Х3 представляет собой Н или D; Х4 представляет собой Т, L, N или V; и X5 представляет собой A, S, Y или T; и/или
область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность X1X2X3X4X5X6 (SEQ ID NO: 1204), где X1 представляет собой F или Y; Х2 представляет собой Q, Y или N; X3 представляет собой G, N, R или Y; X4 представляет собой N, G, E или T; X5 представляет собой S, E, A или G; и X6 представляет собой A, E, I или Q.
55. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 47-54, где TCR или его антигенсвязывающий фрагмент связываются или распознают пептидный эпитоп вируса папилломы человека (HPV) 16 E6 в контексте молекулы МНС, причем пептидный эпитоп представляет собой или содержит E6 (29-38) TIHDIILECV (SEQ ID NO: 233).
56. TCR или антигенсвязывающий фрагмент по любому из вариантов осуществления 47-55, отличающиеся тем, что:
область Vα содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 478, 493, 505, 511, 523, 539, 555, 572, 588, 600, 612, 624, 638, 650, 662 или 679, или CDR3, содержащуюся в аминокислотной последовательности, представленной в любой из SEQ ID NO: 477, 492, 504, 510, 522, 536, 554, 569, 587, 599, 611, 623, 637, 649, 661 или 676; и/или
область Vβ содержит область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 486, 499, 517, 531, 548, 563, 581, 594, 606, 618, 630, 644, 656, 670 или 686, или CDR3, содержащуюся в аминокислотной последовательности, представленной в любой из SEQ ID NO: 483, 498, 516, 530, 545, 560, 578, 593, 605, 617, 629, 643, 655 667 или 685.
57. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 47-56, где область Vα дополнительно содержит:
область 1, определяющую комплементарность (CDR-1), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 136, 161, 165, 537, 570, 142, 171 или 677; и/или
область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 137, 162, 166, 538, 571, 143, 172 или 678.
58. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 47-56, где область Vβ содержит:
область 1, определяющую комплементарность, (CDR-1), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 484, 148, 546, 561, 579, 168, 668 или 154; и/или
область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 485, 149, 547, 562, 580, 169, 669 или 155.
59. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 47-58, отличающиеся тем, что:
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 136, 137 и 478, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 484, 485 и 486, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 161, 162 и 493, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 499, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 165, 166 и 505, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 499, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 161, 162 и 511, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 517, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 136, 137 и 523, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 531, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 537, 538 и 539, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащий аминокислотные последовательности SEQ ID NO: 546, 547 и 548, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 136, 137 и 555, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 561, 562 и 563, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 570, 571 и 572, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 579, 580 и 581, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 136, 137 и 600, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 594, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 136, 137 и 600, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 606, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 136, 137 и 612, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 618, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 136, 137 и 624, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 168, 169 и 630, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 142, 143 и 638, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 561, 562 и 644, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 171, 172 и 650, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 656, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 136, 137 и 662, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 668, 669 и 670, соответственно; или
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 677, 678 и 679, соответственно, и область Vβ содержит CDR-1, CDR-2, и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 686, соответственно.
60. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 47-59, отличающиеся тем, что:
область Vα содержит область 1, определяющую комплементарность, (CDR-1), CDR-2 и CDR-3, соответственно, содержащие аминокислотные последовательности CDR-1, CDR-2 и CDR-3, содержащиеся в аминокислотной последовательности Vα, представленной в любой из SEQ ID NO: 477, 492, 504, 510, 522, 536, 554, 569, 587, 599, 611, 623, 637, 649, 661 или 676; и/или
область Vβ содержит область 1, определяющую комплементарность, (CDR-1), CDR-2 и CDR-3, соответственно, содержащие аминокислотные последовательности CDR-1, CDR-2 и CDR-3, содержащиеся в аминокислотной последовательности Vβ, представленной в любой из SEQ ID NO: 483, 498, 516, 530, 545, 560, 578, 593, 605, 617, 629, 643, 655, 667 или 685.
61. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 47-60, отличающиеся тем, что: области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 477 и 483, соответственно; области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 492 и 498, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 504 и 498, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 510 и 516, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 522 и 530, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 536 и 545, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 554 и 560, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 569 и 578, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 587 и 593, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 599 и 605, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 611 и 617, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 623 и 629, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 637 и 643, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 649 и 655, соответственно; области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 661 и 667, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 676 и 685, соответственно.
62. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 47-61, где альфа-цепь дополнительно содержит константную область альфа (Cα), и/или бета-цепь дополнительно содержит константную область бета (Cβ).
63. TCR или его антигенсвязывающий фрагмент по варианту осуществления 62, где области Cα и Cβ представляют собой константные области мыши.
64. TCR или его антигенсвязывающий фрагмент по варианту осуществления 62 или 63, отличающиеся тем, что:
область Cα содержит аминокислотную последовательность, представленную в SEQ ID NO: 262, 833, 1012, 1014, 1015, 1017, 1018, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности; и/или
область Cβ содержит аминокислотную последовательность, представленную в SEQ ID NO: 263, 1013 или 1016, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности.
65. TCR или его антигенсвязывающий фрагмент по варианту осуществления 62, где области Cα и Cβ представляют собой константные области человека.
66. TCR или его антигенсвязывающий фрагмент по варианту осуществления 62 или 65, отличающиеся тем, что:
область Cα содержит аминокислотную последовательность, представленную в любой из SEQ ID NO: 212, 213, 215, 217, 218, 220 или 524, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности; и/или
область Cβ содержит аминокислотную последовательность, представленную в любой из SEQ ID NO: 214, 216, 631 или 889, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности.
67. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 47-66, отличающиеся тем, что:
а) альфа-цепь содержит:
аминокислотную последовательность, представленную в любой из SEQ ID NO: 473, 488, 500, 506, 518, 532, 550, 565, 583, 595, 607, 619, 633, 645, 657 или 672, последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности; или аминокислотную последовательность, кодируемую нуклеотидной последовательностью, представленной в любой из SEQ ID NO: 389, 430, 1019, 1021, 1023, 1025, 1027, 1029, 1031, 1033, 1035, 1037, 1039, 1041, 1043 или 1045, или нуклеотидной последовательностью, которая имеет с ними по меньшей мере 90% идентичности последовательности; и/или
b) бета-цепь содержит:
аминокислотную последовательность, представленная в любой из SEQ ID NO: 479, 494, 512, 526, 541, 556, 574, 589, 601, 613, 625, 639, 651, 663 или 681, последовательность аминокислот, которая имеет с ними по меньшей мере, 90% идентичности последовательности; или аминокислотную последовательность, кодируемую нуклеотидной последовательностью, представленной в SEQ ID NO: 390, 431, 1020, 1022, 1024, 1026, 1028, 1030, 1032, 1034, 1036, 1038, 1040, 1042, 1044 или 1046, или нуклеотидной последовательностью, которая имеет с ними по меньшей мере 90% идентичности последовательности.
68. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 47-67, отличающиеся тем, что: альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 473 и 479, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 488 и 494, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 500 и 494, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 506 и 512, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 518 и 526, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 532 и 541, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 550 и 556, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 565 и 574, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 583 и 589, соответственно, альфа и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 595 и 601, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 607 и 613, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 619 и 625, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 633 и 639, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 645 и 651, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 657 и 663, соответственно; или альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 672 и 681, соответственно.
69. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 47-68, где TCR или антигенсвязывающий фрагмент содержат одну или более модификаций в α-цепи и/или β-цепи, так что когда TCR или его антигенсвязывающий фрагмент экспрессируются в клетке, частота ошибочного спаривания между α-цепью и β-цепью TCR и эндогенными α-цепью и β-цепью TCR снижается, экспрессия α-цепи и β-цепи TCR увеличивается и/или стабильность α цепи и β-цепи TCR увеличивается, каждая по сравнению с экспрессией в клетке TCR или его антигенсвязывающего фрагмента, не содержащих одну или более модификаций.
70. TCR или его антигенсвязывающий фрагмент по варианту осуществления 69, где одна или более модификаций представляют собой замену, делецию или вставку одной или более аминокислот в области Cα и/или области Cβ.
71. TCR или его антигенсвязывающий фрагмент по варианту осуществления 69 или 70, где одна или более модификаций содержат замену (замены) для введения одного или более остатков цистеина, которые способны образовывать одну или более ненативных дисульфидных связей между альфа-цепью и бета-цепью.
72. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 47-62, 65 и 69-71, содержащие область Cα, содержащую цистеин в положении, соответствующем положению 48, с нумерацией, представленной в SEQ ID NO: 212, 213, 217, 218 или 524 или в положении, соответствующем положению 49, с нумерацией, представленной в SEQ ID NO: 215 или 220; и/или область Cβ, содержащую цистеин в положении, соответствующем положению 57, с нумерацией, представленной в SEQ ID NO: 214 или 216, или в положении, соответствующем положению 58, с нумерацией, представленной в SEQ ID NO: 631 или 889.
73. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 62, 65 и 69-72, отличающиеся тем, что:
область Cα содержит аминокислотную последовательность, представленную в любой из SEQ ID NO: 196, 198, 200, 201, 203 или 525, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности, содержащие один или более остатков цистеина, способных образовывать ненативную дисульфидную связь с бета-цепью; и/или
область Cβ содержит аминокислотную последовательность, представленную в любой из SEQ ID NO: 197, 199, 632 или 890, или последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности, которые содержат один или более остатков цистеина, способных образовывать ненативную дисульфидную связь с альфа-цепью.
74. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 47-73, где TCR или его антигенсвязывающий фрагмент кодируются нуклеотидной последовательностью, которая была оптимизирована по кодонам.
75. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 47-62, 65 и 69-74, отличающиеся тем, что:
а) альфа-цепь содержит:
аминокислотную последовательность, представленную в любой из SEQ ID NO: 474, 489, 501, 507, 519, 533, 551, 566, 584, 596, 608, 620, 634, 646, 658 или 673, последовательность аминокислот, которая имеет с ними по меньшей мере 90% идентичности последовательности; или аминокислотную последовательность, кодируемую нуклеотидной последовательностью, представленной в любой из SEQ ID NO: 1097, 1099, 1101, 1103, 1105, 1107, 1109, 1111, 1113, 1115, 1117, 1119, 1121, 1123, 1125 или 1127, или нуклеотидной последовательностью, которая имеет с ними по меньшей мере 90% идентичности последовательности; и/или
b) бета-цепь содержит:
аминокислотную последовательность, представленную в любой из SEQ ID NO: 480, 495, 513, 527, 542, 557, 575, 590, 602, 614, 626, 640, 652, 664 или 682, последовательность аминокислот, которая имеет с ними по меньшей мере, 90% идентичности последовательности; или аминокислотную последовательность, кодируемую нуклеотидной последовательностью, представленной в SEQ ID NO: 1098, 1100, 1102, 1104, 1106, 1108, 1110, 1112, 1114, 1116, 1118, 1120, 1122, 1124, 1126 или 1128, или нуклеотидной последовательностью, которая имеет с ними по меньшей мере 90% идентичности последовательности.
76. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 47-62, 65 и 69-75, где альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 474 и 482, соответственно; альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 489 и 497, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 501 и 497, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 507 и 515, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 519 и 529, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 533 и 544, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 551 и 559, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 566 и 577, соответственно, альфа и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 584 и 592, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 596 и 604, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 608 и 616, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 620 и 628, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 634 и 642, соответственно, альфа- и бета-цепи включают аминокислотные последовательности SEQ ID NO: 646 и 654, соответственно, альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 658 и 666, соответственно; или альфа- и бета-цепи содержат аминокислотные последовательности SEQ ID NO: 673 и 684, соответственно.
77. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 47-76, где альфа- и/или бета-цепь дополнительно содержит сигнальный пептид.
78. TCR или его антигенсвязывающий фрагмент по варианту осуществления 77, отличающиеся тем, что:
альфа-цепь содержит сигнальный пептид, содержащий аминокислотную последовательность, представленную в любой из SEQ ID NO: 181, 184, 187, 189, 190, 192, 193, 310, 311; и/или
бета-цепь содержит сигнальный пептид, содержащий аминокислотную последовательность, представленную в любой из SEQ ID NO: 182, 185, 186, 188, 191 или 194.
79. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 47-78, которые выделены или очищены или являются рекомбинантными.
80. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 47-79, которые являются человеческими.
81. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 47-80, которые являются моноклональными.
82. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 47-81, где TCR или его антигенсвязывающий фрагмент являются одноцепочечными.
83. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 47-81, где TCR или его антигенсвязывающий фрагмент содержат две цепи.
84. TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 47-83, где антигенспецифичность является по меньшей мере частично CD8-независимой.
85. TCR или антигенсвязывающий фрагмент по любому из вариантов осуществления 47-84, где молекула МНС представляет собой молекулу HLA-A2.
86. Молекула нуклеиновой кислоты, кодирующая TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 47-85 или его альфа- или бета-цепь.
87. Молекула нуклеиновой кислоты по варианту осуществления 86, содержащая нуклеотидную последовательность, кодирующую альфа-цепь, и/или нуклеотидную последовательность, кодирующую бета-цепь, отличающаяся тем, что:
нуклеотидная последовательность, кодирующая альфа-цепь, содержит последовательность, представленную в любой из SEQ ID NO: 389, 430, 1019, 1021, 1023, 1025, 1027, 1029, 1031, 1033, 1035, 1037, 1039, 1041, 1043 или 1045, или нуклеотидную последовательность, которая имеет с ними по меньшей мере 90% идентичности последовательности;
нуклеотидная последовательность, кодирующая бета-цепь, содержит последовательность, представленную в SEQ ID NO: 390, 431, 1020, 1022, 1024, 1026, 1028, 1030, 1032, 1034, 1036, 1038, 1040, 1042, 1044 или 1046, или нуклеотидную последовательность, которая имеет с ними по меньшей мере 90% идентичности последовательности.
88. Молекула нуклеиновой кислоты по варианту осуществления 86, где нуклеотидная последовательность оптимизирована по кодонам.
89. Молекула нуклеиновой кислоты по варианту осуществления 86 или 88, содержащая нуклеотидную последовательность, кодирующую альфа-цепь, и/или нуклеотидную последовательность, кодирующую бета-цепь, отличающаяся тем, что:
нуклеотидная последовательность, кодирующая альфа-цепь, содержит последовательность, представленную в любой из SEQ ID NO: 1097, 1099, 1101, 1103, 1105, 1107, 1109, 1111, 1113, 1115, 1117, 1119, 1121, 1123, 1125 или 1127, или нуклеотидную последовательность, которая имеет с ними по меньшей мере 90% идентичности последовательности;
нуклеотидная последовательность, кодирующая бета-цепь, содержит последовательность, представленную в SEQ ID NO: 1098, 1100, 1102, 1104, 1106, 1108, 1110, 1112, 1114, 1116, 1118, 1120, 1122, 1124, 1126 или 1128, или нуклеотидную последовательность, которая имеет с ними по меньшей мере 90% идентичности последовательности.
90. Молекула нуклеиновой кислоты по любому из вариантов осуществления 86-89, где нуклеотидная последовательность, кодирующая альфа-цепь, и нуклеотидная последовательность, кодирующая бета-цепь, разделены пептидной последовательностью, которая вызывает пропуск рибосомы.
91. Молекула нуклеиновой кислоты по варианту осуществления 90, где пептид, который вызывает пропуск рибосомы, представляет собой пептид P2A или T2A и/или содержит последовательность аминокислот, представленную в SEQ ID NO: 204 или 211.
92. Молекула нуклеиновой кислоты по любому из вариантов осуществления 86-91, содержащая нуклеотидную последовательность, представленную в любой из SEQ ID NO: 432, 433, 434, 435, 436, 437, 438, 439, 440, 441, 442, 443, 444. 445, 446 или 447, или нуклеотидную последовательность, имеющую с ними по меньшей мере 90% идентичности последовательности.
93. Молекула нуклеиновой кислоты по любому из вариантов осуществления 40-46 и 86-92, где молекула нуклеиновой кислоты является синтетической.
94. Молекула нуклеиновой кислоты по любому из вариантов осуществления 40-46 и 86-93, где молекула нуклеиновой кислоты представляет собой кДНК.
95. Полинуклеотид, содержащий:
(а) последовательность нуклеиновой кислоты, кодирующую TCR или его антигенсвязывающую часть по любому из вариантов осуществления 1-39 и 47-85, или молекулу нуклеиновой кислоты по любому из вариантов осуществления 40-46 и 86-94, и
(b) одно или более плеч гомологии, связанных с последовательностью нуклеиновой кислоты, где одно или более плеч гомологии содержат последовательность, гомологичную одной или более областям открытой рамки считывания локуса константной области Т-клеточного рецептора альфа (TRAC).
96. Полинуклеотид, содержащий:
(а) последовательность нуклеиновой кислоты, кодирующую часть Т-клеточного рецептора (TCR), причем указанная последовательность нуклеиновой кислоты кодирует (i) бета-цепь Т-клеточного рецептора (TCRβ), содержащую вариабельную область бета (Vβ) TCR или его антигенсвязывающего фрагмента по любому из вариантов осуществления 1-14, 21-23, 28-39, 47-61, 67-69 и 74-85, и константную область бета (Cβ); и (ii) часть альфа-цепи Т-клеточного рецептора (TCRα), содержащую вариабельную область альфа (Vα) TCR или его антигенсвязывающего фрагмента по любому из вариантов осуществления 1-14, 21-23, 28-39, 47- 61, 67-69 и 74-85, где часть цепи TCRα меньше, чем полноразмерная цепь TCRα, и
(b) одно или более плеч гомологии, связанных с последовательностью нуклеиновой кислоты, где одно или более плеч гомологии содержат последовательность, гомологичную одной или более областям открытой рамки считывания локуса константной области Т-клеточного рецептора альфа (TRAC).
97. Полинуклеотид по варианту осуществления 96, где цепь TCRα содержит константную область альфа (Cα), где по меньшей мере часть указанной Cα кодируется открытой рамкой считывания эндогенного локуса TRAC или его частичной последовательностью, когда TCR или его антигенсвязывающий фрагмент экспрессируется клеткой, в которую был введен полинуклеотид.
98. Полинуклеотид по варианту осуществления 96 или 97, где последовательность нуклеиновой кислоты (а) и одно из одного или более плеч гомологии вместе содержат последовательность нуклеотидов, кодирующую Сα, которая меньше, чем полная длина нативной Сα, где по меньшей мере часть Cα кодируется открытой рамкой считывания эндогенного локуса TRAC или его частичной последовательностью, когда TCR или его антигенсвязывающий фрагмент экспрессируется клеткой, в которую был введен полинуклеотид.
99. Полинуклеотид по любому из вариантов осуществления 96-98, где последовательность нуклеиновой кислоты, кодирующая цепь TCRβ, находится в 5'-области относительно последовательности нуклеиновой кислоты, кодирующей часть цепи TCRα.
100. Полинуклеотид по любому из вариантов осуществления 96-99, где последовательность нуклеиновой кислоты (а) не содержит интрона.
101. Полинуклеотид по любому из вариантов осуществления 96-100, где последовательность нуклеиновой кислоты (а) представляет собой последовательность, которая является экзогенной или гетерологичной относительно открытой рамки считывания эндогенного геномного локуса TRAC Т-клетки, необязательно человеческой Т-клетки.
102. Полинуклеотид по любому из вариантов осуществления 96-101, где последовательность нуклеиновой кислоты (а) находится в рамке с одним или более экзонами или их частичной последовательностью, необязательно с экзоном 1 или его частичной последовательностью, открытой рамки считывания локуса TRAC, содержащейся в одном или более плечах гомологии.
103. Полинуклеотид по любому из вариантов осуществления 97-102, где Cα кодируется открытой рамкой считывания эндогенного локуса TRAC или его частичной последовательностью, а дополнительная часть Cα кодируется последовательностью нуклеиновой кислоты (а), где указанная дополнительная часть Cα меньше чем полная длина нативной Cα.
104. Полинуклеотид по варианту осуществления 103, где дополнительная часть Cα кодируется последовательностью нуклеотидов, начиная с остатка 3 и до остатка 3155 последовательности, представленной в SEQ ID NO: 348, или одним или более ее экзонами или последовательностью, которая имеет по меньшей мере 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичности последовательности с последовательностью нуклеотидов, начиная с остатка 3 и до остатка 3155 последовательности, представленной в SEQ ID NO: 348, или с одним или более ее экзонами, или их частичной последовательностью.
105. Полинуклеотид по варианту осуществления 103 или 104, где дополнительная часть Cα кодируется последовательностью, представленной в SEQ ID NO: 1364, или последовательностью, которая имеет по меньшей мере 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичности последовательности с SEQ ID NO: 1364 или ее частичной последовательностью.
106. Полинуклеотид по любому из вариантов осуществления 103-105, где дополнительная часть области Cα и/или области Cβ, кодируемой последовательностью нуклеиновой кислоты (а), содержит одну или более модификаций, необязательно замену, делецию или вставку одной или более аминокислот по сравнению с нативной областью Cα и/или нативной областью Cβ, необязательно указанные одна или более модификаций вводят один или более остатков цистеина, которые способны образовывать одну или более ненативных дисульфидных связей между альфа-цепью и бета-цепью.
107. Полинуклеотид по любому из вариантов осуществления 95-106, где одно или более плеч гомологии содержат 5'-плечо гомологии и/или 3'-плечо гомологии.
108. Полинуклеотид по любому из вариантов осуществления 95-107, где 5'-плечо гомологии содержит:
а) последовательность, содержащую, или по меньшей мере, содержащую или по меньшей мере 150, 200, 250, 300, 350, 400, 450, 500, 550 или 600 смежных нуклеотидов последовательности, которая имеет по меньшей мере 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичности последовательности с последовательностью, представленной в SEQ ID NO: 1343;
b) последовательность, содержащую, или по меньшей мере, содержащую или, по меньшей мере 150, 200, 250, 300, 350, 400, 450, 500, 550 или 600 смежных нуклеотидов последовательности, представленной в SEQ ID NO: 1343; или
c) последовательность, представленную в SEQ ID NO: 1343.
109. Полинуклеотид по любому из вариантов осуществления 95-108, где 3'-плечо гомологии содержит:
а) последовательность, содержащую или по меньшей мере содержащую или по меньшей мере 150, 200, 250, 300, 350, 400, 450, 500, 550 или 600 смежных нуклеотидов последовательности, которая имеет, по меньшей мере, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичности последовательности с последовательностью, представленной в SEQ ID NO: 1344;
b) последовательность, содержащую или по меньшей мере содержащую или по меньшей мере 150, 200, 250, 300, 350, 400, 450, 500, 550 или 600 смежных нуклеотидов последовательности, представленной в SEQ ID NO: 1344; или
c) последовательность, представленную в SEQ ID NO: 1344.
110. Полинуклеотид, содержащий:
(а) последовательность нуклеиновой кислоты, кодирующую TCR или его антигенсвязывающую часть по любому из вариантов осуществления 1-39 и 47-86, или молекулу нуклеиновой кислоты по любому из вариантов осуществления 40-46 и 86-92, и
(b) одно или более плеч гомологии, связанных с последовательностью нуклеиновой кислоты, где одно или более плеч гомологии содержат последовательность, гомологичную одной или более областям открытой рамки считывания локуса константной области Т-клеточного рецептора бета (TRBC).
111. Полинуклеотид, содержащий:
(а) последовательность нуклеиновой кислоты, кодирующую часть Т-клеточного рецептора (TCR), причем указанная последовательность нуклеиновой кислоты кодирует (i) альфа-цепь Т-клеточного рецептора (TCRα), содержащую вариабельную область альфа (Vα) TCR или его антигенсвязывающего фрагмента по любому из вариантов осуществления 1-14, 21-23, 28-39, 47-61, 67-69 и 74-85 и константную область альфа (Cα); и (ii) часть бета-цепи Т-клеточного рецептора (TCRβ), содержащую вариабельную область бета (Vβ) TCR или его антигенсвязывающего фрагмента по любому из вариантов осуществления 1-14, 21-23, 28-39, 47- 61, 67-69 и 74-85, где часть цепи TCRβ менее, чем полноразмерная цепь TCRβ, и
(b) одно или более плеч гомологии, связанных с последовательностью нуклеиновой кислоты, где одно или более плеч гомологии содержат последовательность, гомологичную одной или более областям открытой рамки считывания локуса константной области Т-клеточного рецептора бета (TRBC).
112. Полинуклеотид по варианту осуществления 111, где цепь TCRβ содержит константную область бета (Cβ), где по меньшей мере часть указанной Cβ кодируется открытой рамкой считывания эндогенного локуса TRBC или его частичной последовательностью, когда TCR или его антигенсвязывающий фрагмент экспрессируются клеткой, в которую был введен полинуклеотид.
113. Полинуклеотид по варианту осуществления 111 или 112, где последовательность нуклеиновой кислоты (а) и одно из одного или более плеч гомологии вместе содержат последовательность нуклеотидов, кодирующую Сβ, которая меньше чем полная длина нативной Сβ, где по меньшей мере, часть Cβ кодируется открытой рамкой считывания эндогенного локуса TRAC или его частичной последовательностью, когда TCR или его антигенсвязывающий фрагмент экспрессируются клеткой, в которую был введен полинуклеотид.
114. Полинуклеотид по любому из вариантов осуществления 111-113, где последовательность нуклеиновой кислоты, кодирующая цепь TCRα, находится в 5'-области относительно последовательности нуклеиновой кислоты, кодирующей часть цепи TCRβ.
115. Полинуклеотид по любому из вариантов осуществления 111-114, где последовательность нуклеиновой кислоты (а) не содержит интрона.
116. Полинуклеотид по любому из вариантов осуществления 111-115, где последовательность нуклеиновой кислоты (а) представляет собой последовательность, которая является экзогенной или гетерологичной по отношению к открытой рамке считывания эндогенного геномного локуса TRBC Т-клетки, необязательно Т-клетки человека.
117. Полинуклеотид по любому из вариантов осуществления 111-116, где последовательность нуклеиновой кислоты (а) находится в рамке с одним или более экзонами или их частичной последовательностью, необязательно экзоном 1 или его частичной последовательностью, открытой рамки считывания локуса TRBC, содержащейся в одном или более плечах гомологии.
118. Полинуклеотид по любому из вариантов осуществления 111-117, где часть Cβ кодируется открытой рамкой считывания эндогенного локуса TRBC или его частичной последовательностью, а дополнительная часть Cβ кодируется последовательностью нуклеиновой кислоты (а), где указанная дополнительная часть Cβ меньше чем полная длина нативной Cβ.
119. Полинуклеотид по варианту осуществления 118, где дополнительная часть Cβ кодируется:
последовательностью нуклеотидов, начиная с остатка 3 и до остатка 1445 последовательности, представленной в SEQ ID NO: 349, или одним или более ее экзонами, или последовательностью, которая имеет, по меньшей мере, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичности последовательности с последовательностью нуклеотидов, начиная с остатка 3 и до остатка 1445 последовательности, представленной в SEQ ID NO: 349, или одним или более ее экзонами, или их частичной последовательностью; или
последовательностью нуклеотидов, начиная с остатка 3 и до остатка 1486 последовательности, представленной в SEQ ID NO: 1047, или одним или более ее экзонами, или последовательностью, которая имеет по меньшей мере 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичности последовательности с последовательностью нуклеотидов, начиная с остатка 3 и до остатка 1486 последовательности, представленной в SEQ ID NO: 1047, или одним или более ее экзонами, или их частичной последовательностью;
120. Полинуклеотид по варианту осуществления 118 или 119, где дополнительная часть области Cβ и/или Cα, кодируемая последовательностью нуклеиновой кислоты (а), содержит одну или более модификаций, необязательно замену, делецию или вставку одной или более аминокислот по сравнению с нативной областью Cβ и/или нативной областью Cα, причем необязательно указанные одна или более модификаций вводят один или более остатков цистеина, которые способны образовывать одну или более ненативных дисульфидных связей между альфа-цепью и бета-цепью.
121. Полинуклеотид по любому из вариантов осуществления 110-120, где одно или более плеч гомологии содержат 5'-плечо гомологии и/или 3'-плечо гомологии.
122. Полинуклеотид по любому из вариантов осуществления 95-121, где последовательность нуклеиновой кислоты (а) содержит один или более полицистронных элементов.
123. Полинуклеотид по варианту осуществления 122, где полистронный элемент(элементы) расположен между последовательностью нуклеиновой кислоты, кодирующей TCRα или ее часть, и последовательностью нуклеиновой кислоты, кодирующей TCRβ или ее часть.
124. Полинуклеотид по варианту осуществления 122 или 123, где один или более полицистронных элементов находятся в 5'-области относительно последовательности нуклеиновой кислоты, кодирующей TCR, или часть TCR, или молекулы нуклеиновой кислоты, кодирующей TCR.
125. Полинуклеотид по любому из вариантов осуществления 122-124, где один или более полицистронных элементов представляют собой или содержат последовательность пропуска рибосомы, необязательно, где последовательность пропуска рибосомы представляет собой элемент T2A, P2A, E2A или F2A.
126. Полинуклеотид по любому из вариантов осуществления 95-125, где последовательность нуклеиновой кислоты (а) содержит один или более гетерологичных или регуляторных контрольных элементов, функционально связанных с контролем экспрессии TCR, когда он экспрессируется клеткой, в которую был введен полинуклеотид.
127. Полинуклеотид по варианту осуществления 126, где один или более гетерологичных регуляторных или контрольных элементов содержат промотор, энхансер, интрон, сигнал полиаденилирования, консенсусную последовательность Козака, акцепторную последовательность сплайсинга и/или донорную последовательность сплайсинга.
128. Полинуклеотид по варианту осуществления 126 или 127, где гетерологичный регуляторный или контрольный элемент содержат гетерологичный промотор, необязательно промотор фактора элонгации 1 альфа (EF1α) человека или промотор MND, или его вариант.
129. Полинуклеотид по любому из вариантов осуществления 95-128, который представляет собой линейный полинуклеотид, необязательно двухцепочечный полинуклеотид или одноцепочечный полинуклеотид.
130. Вектор, содержащий молекулу нуклеиновой кислоты по любому из вариантов осуществления 40-46 и 86-94 или полинуклеотид по любому из вариантов осуществления 95-129.
131. Вектор по варианту осуществления 130, где вектор представляет собой экспрессирующий вектор.
132. Вектор по варианту осуществления 130 или 131, где вектор представляет собой вирусный вектор.
133. Вектор по варианту осуществления 132, где вирусный вектор представляет собой ретровирусный вектор.
134. Вектор по варианту осуществления 132 или 133, где вирусный вектор представляет сбой лентивирусный вектор.
135. Вектор по варианту осуществления 134, где лентивирусный вектор получен из HIV-1.
136. Вектор по варианту осуществления 132 или 133, где вирусный вектор представляет собой гаммаретровирусный вектор.
137. Вектор по варианту осуществления 132, где вирусный вектор представляет собой вектор AAV.
138 Вектор по варианту осуществления 137, где вектор AAV выбран из векторов AAV1, AAV2, AAV3, AAV4, AAV5, AAV6, AAV7 или AAV8.
139. Сконструированная клетка, содержащая молекулу нуклеиновой кислоты по любому из вариантов осуществления 40-46 и 86-94, полинуклеотид по любому из вариантов осуществления 95-129 или вектор по любому из вариантов осуществления 130-138.
140. Сконструированная клетка, содержащая TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 1-39 и 47-85, необязательно рекомбинантный TCR или антигенсвязывающий фрагмент по любому из вариантов осуществления 1-39 и 47-85.
141. Сконструированная клетка по варианту осуществления 139 или 140, содержащая генетическое нарушение гена константной области эндогенного Т-клеточного рецептора альфа (TRAC) и/или гена константной области Т-клеточного рецептора бета (TRBC).
142. Сконструированная клетка по варианту осуществления 141, где ген TRBC представляет собой один или оба из гена константной области 1 Т-клеточного рецептора бета (TRBC1) или гена константной области 2 Т-клеточного рецептора бета (TRBC2).
143. Сконструированная клетка по варианту осуществления 141 или 142, где сконструированная клетка не содержит непрерывного гена TRAC и/или TRBC; не содержит ген TRAC и/или TRBC; не содержит функционального гена TRAC и/или TRBC; и/или не экспрессирует, не экспрессирует на детектируемом уровне или экспрессирует на уровне менее чем 20%, 15%, 10%, 5%, 4%, 3%, 2% или 1% от уровня генного продукта эндогенного TRAC или TRBC дикого типа.
144. Сконструированная клетка, содержащая TCR или его антигенсвязывающий фрагмент, необязательно рекомбинантный TCR или его антигенсвязывающий фрагмент, где:
(1) клетка содержит генетическое нарушение гена константной области Т-клеточного рецептора альфа (TRAC) и/или гена константной области Т-клеточного рецептора бета (TRBC) и/или не экспрессирует, или не экспрессирует на детектируемом уровне или экспрессирует на уровне менее чем 20%, 15%, 10%, 5%, 4%, 3%, 2% или 1% от уровня генного продукта эндогенного TRAC или TRBC дикого типа; и
(2) TCR или его антигенсвязывающий фрагмент или рекомбинантный TCR или его антигенсвязывающий фрагмент включают TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 1-39 и 47-85, необязательно рекомбинантный TCR или антигенсвязывающий фрагмент по любому из вариантов осуществления 1-39 и 47-85.
145. Сконструированная клетка, содержащая TCR или его антигенсвязывающий фрагмент, необязательно рекомбинантный TCR или его антигенсвязывающий фрагмент, где:
(1) клетка содержит генетическое нарушение гена константной области Т-клеточного рецептора альфа (TRAC) и/или гена константной области Т-клеточного рецептора бета (TRBC) и/или не экспрессирует, или не экспрессирует на детектируемом уровне или экспрессирует на уровне менее чем 20%, 15%, 10%, 5%, 4%, 3%, 2% или 1% от уровня генного продукта эндогенного TRAC или TRBC дикого типа; и
(2) TCR или его антигенсвязывающий фрагмент или рекомбинантный TCR или его антигенсвязывающий фрагмент содержат:
(а) вариабельную область альфа (Vα), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 117, 119 или 295, или аминокислотную последовательность, которая имеет с ними по меньшей мере 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, или 99% идентичности последовательности, и вариабельную область бета (Vβ), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 118, 120 или 296, или аминокислотную последовательность, которая имеет с ними по меньшей мере 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, или 99% идентичности последовательности; и/или
(b) область Vα, содержащую область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 153, 159 или 301, или CDR3, содержащуюся в аминокислотной последовательности, представленной в любой из SEQ ID NO: 117, 119 или 295, и область Vβ, содержащую область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 156 или 160, или CDR3, содержащуюся в аминокислотной последовательности, представленной в любой из SEQ ID NO: 118, 120 или 296.
146. Сконструированная клетка по варианту осуществления 145, отличающаяся тем, что:
область Vα дополнительно содержит область 1, определяющую комплементарность, (CDR-1), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 151 или 157; и/или область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 152 или 158; и/или
область Vβ дополнительно содержит область 1, определяющую комплементарность, (CDR-1), содержащую аминокислотную последовательность, представленную в SEQ ID NO: 154; и/или область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность, представленную в SEQ ID NO: 155.
147. Сконструированная клетка по варианту осуществления 145 или 146, где область Vα и область Vβ содержат:
CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 151, 152 и 153, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 156, соответственно;
CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 157, 158 и 159, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 160, соответственно; или
CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 151, 152 и 301, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 156, соответственно.
148. Сконструированная клетка по варианту осуществления 145, отличающаяся тем, что:
область Vα содержит область 1, определяющую комплементарность, (CDR-1), CDR-2 и CDR-3, соответственно, содержащие аминокислотные последовательности CDR-1, CDR-2 и CDR-3, содержащиеся в аминокислотной последовательности области Vα, представленной в любой из SEQ ID NO: 117, 119 или 295; и/или
область Vβ содержит область 1, определяющую комплементарность, (CDR-1), CDR-2 и CDR-3, соответственно, содержащие аминокислотные последовательности CDR-1, CDR-2 и CDR-3, содержащиеся в аминокислотной последовательности области Vβ, представленной в любой из SEQ ID NO: 118, 120 или 296.
149. Сконструированная клетка по любому из вариантов осуществления 145-148:
области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 117 и либо 118, либо 296, соответственно;
области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 119 и 120, соответственно; или
области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 295 и либо 118, либо 296, соответственно.
150. Сконструированная клетка по любому из вариантов осуществления 145-149, где TCR или его антигенсвязывающий фрагмент связываются или распознают пептидный эпитоп вируса папилломы человека (HPV) 16 E7 в контексте молекулы МНС, причем пептидный эпитоп представляет собой или содержит E7 (11-19) YMLDLQPET (SEQ ID NO: 236).
151. Сконструированная клетка, содержащая TCR или его антигенсвязывающий фрагмент, необязательно рекомбинантный TCR или его антигенсвязывающий фрагмент, где:
(1) клетка содержит генетическое нарушение гена константной области Т-клеточного рецептора альфа (TRAC) и/или гена константной области Т-клеточного рецептора бета (TRBC), и/или не экспрессирует, или не экспрессирует на детектируемом уровне или экспрессирует на уровне менее чем 20%, 15%, 10%, 5%, 4%, 3%, 2% или 1% от уровня генного продукта эндогенного TRAC или TRBC дикого типа; и
(2) TCR или его антигенсвязывающий фрагмент или рекомбинантный TCR или его антигенсвязывающий фрагмент содержат:
(a) вариабельную область альфа (Vα), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 111, 113, 115, 121, 123, 125, 297 или 299, или аминокислотную последовательность, которая имеет с ними в по меньшей мере 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% идентичности последовательности; и/или область Vβ, содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: SEQ ID NO: 112, 114, 116, 122, 124, 126, 298 или 300, или аминокислотную последовательность, которая имеет с ними по меньшей мере 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% идентичности последовательности; и/или
(b) область Vα, содержащую область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 138, 144, 147, 163, 167, 173, 304 или 308, или CDR3, содержащуюся в аминокислотной последовательности, представленной в любой из SEQ ID NO: 111, 113, 115, 121, 123, 125, 297 или 299, и область Vβ, содержащую область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 141, 146, 150, 164, 170, 174, 305 или 309, или CDR3, содержащуюся в аминокислотной последовательности, представленной в любой из SEQ ID NO: 112, 114, 116, 122, 124, 126, 298 или 300.
152. Сконструированная клетка по варианту осуществления 151, отличающаяся тем, что:
область Vα дополнительно содержит область 1, определяющую комплементарность, (CDR-1), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 136, 142, 161, 165, 171, 302 или 306, или CDR-1, содержащуюся в аминокислотной последовательности, представленной в любой из SEQ ID NO: 111, 113, 115, 121, 123, 125, 297 или 299; и/или область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 137, 143, 162, 166, 172, 303 или 307, или CDR-2, содержащуюся в аминокислотной последовательности, представленной в любой из SEQ ID NO: 111, 113, 115, 121, 123, 125, 297 или 299; и/или
область Vβ дополнительно содержит область 1, определяющую комплементарность, (CDR-1), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 139, 145, 148, 168, или CDR-1, содержащуюся в аминокислотной последовательности, представленную в любой из SEQ ID NO: 112, 114, 116, 122, 124, 126, 298 или 300; и/или область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 140, 149 или 169, или CDR-2, содержащуюся в аминокислотной последовательности, представленной в любой из SEQ ID NO: 112, 114, 116, 122, 124, 126, 298 или 300.
153. Сконструированная клетка по варианту осуществления 151 или 152, отличающаяся тем, что
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 136, 137 и 138, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 139, 140 и 141, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 142, 143 и 144, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 145, 140 и 146, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 136, 137 и 147, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 150, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 161, 162 и 163, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 164, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 165, 166 и 167, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 168, 169 и 170, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 171, 172 и 173, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 174, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 302, 303 и 304, соответственно, а область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 139, 140 и 305, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 306, 307 и 308, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 309, соответственно.
154. Сконструированная клетка по любому из вариантов осуществления 151-153, отличающаяся тем, что:
область Vα содержит область 1, определяющую комплементарность, (CDR-1), CDR-2 и CDR-3, соответственно, содержащие аминокислотные последовательности CDR-1, CDR-2 и CDR-3, содержащиеся в аминокислотной последовательности области Vα, представленной в любой из SEQ ID NO: 111, 113, 115, 121, 123, 125, 297 или 299; и/или
область Vβ содержит область 1, определяющую комплементарность, (CDR-1), CDR-2 и CDR-3, соответственно содержащие аминокислотные последовательности CDR-1, CDR-2 и CDR-3, содержащиеся в аминокислотной последовательности области Vβ, представленной в любой из SEQ ID NO: 112, 114, 116, 122, 124, 126, 298 или 300.
155. Сконструированная клетка по любому из вариантов осуществления 151-154: области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 111 и 112, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 113 и 114, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 115 и 116, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 121 и 122, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 123 и 124, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 125 и 126, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 297 и 298, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 299 и 300, соответственно.
156. Сконструированная клетка по любому из вариантов осуществления 151-155, где TCR или его антигенсвязывающий фрагмент связываются или распознают пептидный эпитоп вируса папилломы человека (HPV) 16 E6 в контексте молекулы МНС, причем пептидный эпитоп представляет собой или содержит E6 (29-38) TIHDIILECV (SEQ ID NO: 233).
157. Сконструированная клетка по любому из вариантов осуществления 145-156, где альфа-цепь дополнительно содержит константную область альфа (Cα), и/или бета-цепь дополнительно содержит константную область бета (Cβ).
158. Сконструированная клетка по любому из вариантов осуществления 143-157, где генный продукт представляет собой мРНК или белок, кодируемый геном TRAC или TRBC.
159. Сконструированная клетка по любому из вариантов осуществления 141-158, где генетическое нарушение включает мутацию или делецию в области гена TRAC или TRBC, которые находятся в кодирующей области, необязательно ранней кодирующей области гена, в пределах экзона 1 гена, в кодирующей области в пределах 500, 400, 300, 200, 100 или 50 пар оснований от старт-кодона гена, в последовательности сайта-мишени, которая комплементарна нацеливающему сайту нацеливающего домена гид-РНК (гРНК), имеющего последовательность, выбранную из любой из SEQ ID NO: 1053 и 1259-1315, и/или с которой специфически гибридизуется нацеливающий домен, имеющий последовательность, выбранную из SEQ ID NO: 1053 и 1259-1315,, и/или в последовательности сайта-мишени, которая комплементарна нацеливающему сайту нацеливающего домена гРНК, имеющего последовательность, выбранную из любой из SEQ ID NO: 1048 и 1229-1258, и/или с которой специфически гибридизуется нацеливающий домен, имеющий последовательность, выбранную из SEQ ID NO: 1048 и 1229-1258.
160. Сконструированная клетка по любому из вариантов осуществления 141-159, где на генетическое нарушение влияет один или более агентов, которые содержат (a) по меньшей мере одну гРНК, имеющую нацеливающий домен, который комплементарен домену-мишени гена TRAC и/или гена TRBC или (b) по меньшей мере одну нуклеиновую кислоту, кодирующую по меньшей мере одну гРНК.
161. Сконструированная клетка по любому из вариантов осуществления 141-160, где один или более агентов содержат, по меньшей мере, один комплекс молекулы Cas9 и, по меньшей мере, одну гРНК, имеющую нацеливающий домен, который комплементарен домену-мишени гена TRAC и/или гена TRBC.
162. Сконструированная клетка по любому из вариантов осуществления 160 или 161, где по меньшей мере одна гРНК содержит нацеливающий домен, который комплементарен домену-мишени гена TRAC, причем указанный нацеливающий домен содержит последовательность, выбранную из UCUCUCAGCUGGUACACGGC (SEQ ID NO:1229), UGGAUUUAGAGUCUCUCAGC (SEQ ID NO:1230), ACACGGCAGGGUCAGGGUUC (SEQ ID NO:1231), GAGAAUCAAAAUCGGUGAAU (SEQ ID NO:1048), GCUGGUACACGGCAGGGUCA (SEQ ID NO:1232), CUCAGCUGGUACACGGC (SEQ ID NO:1233), UGGUACACGGCAGGGUC (SEQ ID NO:1234), GCUAGACAUGAGGUCUA (SEQ ID NO:1235), GUCAGAUUUGUUGCUCC (SEQ ID NO:1236), UCAGCUGGUACACGGCA (SEQ ID NO:1237), GCAGACAGACUUGUCAC (SEQ ID NO:1238), GGUACACGGCAGGGUCA (SEQ ID NO:1239), CUUCAAGAGCAACAGUGCUG (SEQ ID NO:1240), AGAGCAACAGUGCUGUGGCC (SEQ ID NO:1241), AAAGUCAGAUUUGUUGCUCC (SEQ ID NO:1242), ACAAAACUGUGCUAGACAUG (SEQ ID NO:1243), AAACUGUGCUAGACAUG (SEQ ID NO:1244), UGUGCUAGACAUGAGGUCUA (SEQ ID NO:1245), GGCUGGGGAAGAAGGUGUCUUC (SEQ ID NO:1246), GCUGGGGAAGAAGGUGUCUUC (SEQ ID NO:1247), GGGGAAGAAGGUGUCUUC (SEQ ID NO:1248), GUUUUGUCUGUGAUAUACACAU (SEQ ID NO:1249), GGCAGACAGACUUGUCACUGGAUU (SEQ ID NO:1250), GCAGACAGACUUGUCACUGGAUU (SEQ ID NO:1251), GACAGACUUGUCACUGGAUU (SEQ ID NO:1252), GUGAAUAGGCAGACAGACUUGUCA (SEQ ID NO:1253), GAAUAGGCAGACAGACUUGUCA (SEQ ID NO:1254), GAGUCUCUCAGCUGGUACACGG (SEQ ID NO:1255), GUCUCUCAGCUGGUACACGG (SEQ ID NO:1256), GGUACACGGCAGGGUCAGGGUU (SEQ ID NO:1257), и GUACACGGCAGGGUCAGGGUU (SEQ ID NO:1258).
163. Сконструированная клетка по любому из вариантов осуществления 160-162, где гРНК содержит нацеливающий домен, который комплементарен домену-мишени гена TRBC, необязательно в одном или обоих генах TRBC1 и TRBC2, причем указанный нацеливающий домен-мишень содержит последовательность, выбранную из CACCCAGAUCGUCAGCGCCG (SEQ ID NO:1259), CAAACACAGCGACCUCGGGU (SEQ ID NO:1260), UGACGAGUGGACCCAGGAUA (SEQ ID NO:1261), GGCUCUCGGAGAAUGACGAG (SEQ ID NO:1262), GGCCUCGGCGCUGACGAUCU (SEQ ID NO:1053), GAAAAACGUGUUCCCACCCG (SEQ ID NO:1263), AUGACGAGUGGACCCAGGAU (SEQ ID NO:1264), AGUCCAGUUCUACGGGCUCU (SEQ ID NO:1265), CGCUGUCAAGUCCAGUUCUA (SEQ ID NO:1266), AUCGUCAGCGCCGAGGCCUG (SEQ ID NO:1267), UCAAACACAGCGACCUCGGG (SEQ ID NO:1268), CGUAGAACUGGACUUGACAG (SEQ ID NO:1269), AGGCCUCGGCGCUGACGAUC (SEQ ID NO:1270), UGACAGCGGAAGUGGUUGCG (SEQ ID NO:1271), UUGACAGCGGAAGUGGUUGC (SEQ ID NO:1272), UCUCCGAGAGCCCGUAGAAC (SEQ ID NO:1273), CGGGUGGGAACACGUUUUUC (SEQ ID NO:1274), GACAGGUUUGGCCCUAUCCU (SEQ ID NO:1275), GAUCGUCAGCGCCGAGGCCU (SEQ ID NO:1276), GGCUCAAACACAGCGACCUC (SEQ ID NO:1277), UGAGGGUCUCGGCCACCUUC (SEQ ID NO:1278), AGGCUUCUACCCCGACCACG (SEQ ID NO:1279), CCGACCACGUGGAGCUGAGC (SEQ ID NO:1280), UGACAGGUUUGGCCCUAUCC (SEQ ID NO:1281), CUUGACAGCGGAAGUGGUUG (SEQ ID NO:1282), AGAUCGUCAGCGCCGAGGCC (SEQ ID NO:1283), GCGCUGACGAUCUGGGUGAC (SEQ ID NO:1284), UGAGGGCGGGCUGCUCCUUG (SEQ ID NO:1285), GUUGCGGGGGUUCUGCCAGA (SEQ ID NO:1286), AGCUCAGCUCCACGUGGUCG (SEQ ID NO:1287), GCGGCUGCUCAGGCAGUAUC (SEQ ID NO:1288), GCGGGGGUUCUGCCAGAAGG (SEQ ID NO:1289), UGGCUCAAACACAGCGACCU (SEQ ID NO:1290), ACUGGACUUGACAGCGGAAG (SEQ ID NO:1291), GACAGCGGAAGUGGUUGCGG (SEQ ID NO:1292), GCUGUCAAGUCCAGUUCUAC (SEQ ID NO:1293), GUAUCUGGAGUCAUUGAGGG (SEQ ID NO:1294), CUCGGCGCUGACGAUCU (SEQ ID NO:1295), CCUCGGCGCUGACGAUC (SEQ ID NO:1296), CCGAGAGCCCGUAGAAC (SEQ ID NO:1297), CCAGAUCGUCAGCGCCG (SEQ ID NO:1298), GAAUGACGAGUGGACCC (SEQ ID NO:1299), GGGUGACAGGUUUGGCCCUAUC (SEQ ID NO:1300), GGUGACAGGUUUGGCCCUAUC (SEQ ID NO:1301), GUGACAGGUUUGGCCCUAUC (SEQ ID NO:1302), GACAGGUUUGGCCCUAUC (SEQ ID NO:1303), GAUACUGCCUGAGCAGCCGCCU (SEQ ID NO:1304), GACCACGUGGAGCUGAGCUGGUGG (SEQ ID NO:1305), GUGGAGCUGAGCUGGUGG (SEQ ID NO:1306), GGGCGGGCUGCUCCUUGAGGGGCU (SEQ ID NO:1307), GGCGGGCUGCUCCUUGAGGGGCU (SEQ ID NO:1308), GCGGGCUGCUCCUUGAGGGGCU (SEQ ID NO:1309), GGGCUGCUCCUUGAGGGGCU (SEQ ID NO:1310), GGCUGCUCCUUGAGGGGCU (SEQ ID NO:1311), GCUGCUCCUUGAGGGGCU (SEQ ID NO:1312), GGUGAAUGGGAAGGAGGUGCACAG (SEQ ID NO:1313), GUGAAUGGGAAGGAGGUGCACAG (SEQ ID NO:1314), и GAAUGGGAAGGAGGUGCACAG (SEQ ID NO:1315).
164. Сконструированная клетка по любому из вариантов осуществления 141-163, отличающаяся тем, что:
сконструированная клетка содержит генетическое нарушение гена константной области Т-клеточного рецептора альфа (TRAC) и гена константной области Т-клеточного рецептора бета (TRBC); и/или
один или более агентов содержат агент, содержащий, по меньшей мере, одну гРНК, имеющую нацеливающий домен, который комплементарен домену-мишени гена TRAC, и агент, содержащий, по меньшей мере, одну гРНК, имеющую нацеливающий домен, который комплементарен домену-мишени гена TRBC, необязательно одного или обоих, гена TRBC1 и гена TRBC2.
165. Сконструированная клетка по любому из вариантов осуществления 159-164, где нацеливающий домен содержит последовательность, комплементарную домену-мишени гена TRAC, и нацеливающий домен содержит последовательность GAGAAUCAAAAUCGGUGAAU (SEQ ID NO: 1048)
166. Сконструированная клетка по любому из вариантов осуществления 159-165, где нацеливающий домен содержит последовательность, комплементарную домену-мишени гена TRBC, и нацеливающий домен содержит последовательность GGCCUCGGCGCUGACGAUCU (SEQ ID NO: 1053).
167. Сконструированная клетка по любому из вариантов осуществления 159-166, где гРНК дополнительно содержит первый домен комплементарности, второй домен комплементарности, который комплементарен первому домену комплементарности, проксимальный домен и, необязательно, хвостовой домен.
168. Сконструированная клетка по варианту осуществления 167, где первый домен комплементарности и второй домен комплементарности соединены связывающим доменом.
169. Сконструированная клетка по варианту осуществления 167 или 168, где гид-РНК содержит 3'-поли-А-хвост и 5'-Cap-аналог (Anti-Reverse Cap Analog) (ARCA).
170. Сконструированная клетка по любому из вариантов осуществления 161-169, где молекула Cas9 является ферментативно активной Cas9.
171. Сконструированная клетка по любому из вариантов осуществления 161-170, где молекула Cas9 представляет собой молекулу Cas9 S. aureus.
172. Сконструированная клетка по любому из вариантов осуществления 161-171, где молекула Cas9 представляет собой Cas9 S. pyogenes.
173. Сконструированная клетка по любому из вариантов осуществления 141-172, где сконструированная клетка содержит генетическое нарушение локуса константной области Т-клеточного рецептора альфа (TRAC).
174. Сконструированная клетка по любому из вариантов осуществления 141-173, где эндогенный локус TRAC дополнительно модифицируется путем интеграции последовательности нуклеиновой кислоты, кодирующей TCR или его антигенсвязывающий фрагмент, в локус TRAC, необязательно через HDR.
175. Сконструированная клетка по любому из вариантов осуществления 141-173, где эндогенный локус TRAC дополнительно модифицируется путем интеграции трансгенной последовательности, кодирующей часть TCR или его антигенсвязывающего фрагмента, необязательно посредством направляемой гомологией репарации (HDR).
176. Сконструированная клетка, содержащая модифицированный локус TRAC, кодирующий TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 1-39 и 47-85.
177. Сконструированная клетка, содержащая модифицированный локус TRAC, где эндогенный локус TRAC модифицируется путем интеграции трансгенной последовательности, кодирующей часть TCR, причем указанная трансгенная последовательность кодирует (i) бета-цепь T-клеточного рецептора (TCRβ), содержащую вариабельную область бета (Vβ) TCR или его антигенсвязывающего фрагмента по любому из вариантов осуществления 1-14, 21-23, 28-39, 47-61, 67-69 и 74-85, и константную область бета (Cβ); и (ii) часть альфа-цепи Т-клеточного рецептора (TCRα), содержащую вариабельную область альфа (Vα) TCR или его антигенсвязывающего фрагмента по любому из вариантов осуществления 1-14, 21-23, 28-39, 47- 61, 67-69 и 74-85, где по меньшей мере часть константной области альфа (Cα) TCR кодируется открытой рамкой считывания эндогенного локуса TRAC или его частичной последовательностью.
178. Сконструированная клетка по любому из вариантов осуществления 174-176, где TCR или его антигенсвязывающий фрагмент содержит Cα, причем по меньшей мере, часть указанной Cα кодируется открытой рамкой считывания или ее частичной последовательностью эндогенного локуса TRAC.
179. Сконструированная клетка по любому из вариантов осуществления 174-178, где модифицированный локус TRAC содержит слияние в рамке (i) трансгенной последовательности, кодирующей часть TCR, и (ii) открытой рамки считывания или ее частичной последовательности эндогенного локуса TRAC.
180. Сконструированная клетка по любому из вариантов осуществления 175, 177 и 179, где трансгенная последовательность не содержит последовательности, кодирующей 3'-UTR или интрон.
181. Сконструированная клетка по любому из вариантов осуществления 177-180, где открытая рамка считывания или ее частичная последовательность содержит 3'-UTR эндогенного локуса TRAC.
182. Сконструированная клетка по любому из вариантов осуществления 176, 177 и 179-181, где трансгенная последовательность интегрирована в 3'-области относительно крайнего 5'-нуклеотида экзона 1 и в 5'-области относительно крайнего 3'-нуклеотида экзона 1 открытой рамки считывания эндогенного локуса TRAC.
183. Сконструированная клетка по любому из вариантов осуществления 177-182, где по меньшей мере часть Cα кодируется по меньшей мере экзонами 2-4 открытой рамки считывания эндогенного локуса TRAC.
184. Сконструированная клетка по любому из вариантов осуществления 177-183, отличающаяся тем, что, по меньшей мере, часть Cα кодируется, по меньшей мере, частью экзона 1 и экзонов 2-4 открытой рамки считывания эндогенного локуса TRAC.
185. Сконструированная клетка по любому из вариантов осуществления 175, 177 и 179-184, где трансгенная последовательность кодирует бета-цепь Т-клеточного рецептора (TCRβ) и/или вариабельную область TCR альфа (Vα).
186. Сконструированная клетка по любому из вариантов осуществления 173-185, дополнительно содержащая генетическое нарушение локуса константной области Т-клеточного рецептора бета (TRBC), необязательно локуса TRBC1 или TRBC2.
187. Сконструированная клетка по любому из вариантов осуществления 141-173, где сконструированная клетка содержит генетическое нарушение локуса константной области Т-клеточного рецептора бета (TRBC).
188. Сконструированная клетка по любому из вариантов осуществления 141-173, где эндогенный локус TRBC дополнительно модифицируется путем интеграции последовательности нуклеиновой кислоты, кодирующей TCR или его антигенсвязывающий фрагмент, в локус TRBC, необязательно посредством HDR.
189. Сконструированная клетка по любому из вариантов осуществления 141-173, где эндогенный локус TRBC дополнительно модифицируется путем интеграции трансгенной последовательности, кодирующей часть TCR или его антигенсвязывающего фрагмента, необязательно посредством репарации, направляемой гомологией, (HDR).
190. Сконструированная клетка, содержащая модифицированный локус TRBC, кодирующий TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 1-39 и 47-85.
191. Сконструированная клетка, содержащая модифицированный локус TRBC, где эндогенный локус TRBC модифицируется путем интеграции трансгенной последовательности, кодирующей часть TCR, причем указанная трансгенная последовательность кодирует (i) альфа-цепь Т-клеточного рецептора (TCRα), содержащую вариабельную область альфа (Vα) TCR или его антигенсвязывающего фрагмента по любому из вариантов осуществления 1-14, 21-23, 28-39, 47-61, 67-69 и 74-85, и константную область альфа (Cα); и (ii) часть бета-цепи Т-клеточного рецептора (TCRβ), содержащую вариабельную область бета (Vβ) TCR или его антигенсвязывающего фрагмента по любому из вариантов осуществления 1-14, 21-23, 28-39, 47- 61, 67-69 и 74-85, где по меньшей мере часть константной области бета (Cβ) TCR кодируется открытой рамкой считывания эндогенного локуса TRBC или его частичной последовательностью.
192. Сконструированная клетка по любому из вариантов осуществления 188-190, где TCR или его антигенсвязывающий фрагмент содержат Cβ, причем по меньшей мере, часть указанной Cβ кодируется открытой рамкой считывания или ее частичной последовательностью эндогенного локуса TRBC.
193. Сконструированная клетка по любому из вариантов осуществления 188-192, где модифицированный локус TRBC содержит слияние в рамке (i) трансгенной последовательности, кодирующей часть TCR, и (ii) открытой рамки считывания или ее частичной последовательности эндогенного локуса TRBC.
194. Сконструированная клетка по любому из вариантов осуществления 189, 191 и 193, где трансгенная последовательность не содержит последовательности, кодирующей 3'-UTR или интрон.
195. Сконструированная клетка по любому из вариантов осуществления 192-194, где открытая рамка считывания или ее частичная последовательность содержат 3'-UTR эндогенного локуса TRBC.
196. Сконструированная клетка по любому из вариантов осуществления 189, 191 и 193-195, где трансгенная последовательность интегрирована в 3'-области относительно крайнего 5'-нуклеотида экзона 1 и в 5'-области относительно крайнего 3'-нуклеотида экзона 1 открытой рамки считывания эндогенного локуса TRBC.
197. Сконструированная клетка по любому из вариантов осуществления 191-196, отличающаяся тем, что, по меньшей мере, часть Cβ кодируется, по меньшей мере, экзонами 2-4 открытой рамки считывания эндогенного локуса TRBC.
198. Сконструированная клетка по любому из вариантов осуществления 191-197, отличающаяся тем, что, по меньшей мере, часть Cβ кодируется, по меньшей мере, частью экзона 1 и экзонов 2-4 открытой рамки считывания эндогенного локуса TRBC.
199. Сконструированная клетка по любому из вариантов осуществления 189, 191 и 193-198, где трансгенная последовательность кодирует альфа-цепь Т-клеточного рецептора (TCRα) и/или вариабельную область TCR бета (Vβ).
200. Сконструированная клетка по любому из вариантов осуществления 187-199, где локус TRBC представляет собой один или оба из локуса константной области 1 Т-клеточного рецептора бета (TRBC1) или константной области 2 Т-клеточного рецептора бета (TRBC2).
201. Сконструированная клетка по любому из вариантов осуществления 188-200, дополнительно содержащая генетическое нарушение локуса константной области Т-клеточного рецептора альфа (TRAC).
202. Сконструированная клетка по любому из вариантов осуществления 174-186 и 188-201, где трансгенная последовательность или последовательность нуклеиновой кислоты, кодирующая TCR или его антигенсвязывающий фрагмент, содержит один или более полицистронных элементов.
203. Сконструированная клетка по варианту осуществления 202, где один или более полицистронных элементов находятся в 5'-области от трансгенной последовательности или последовательности нуклеиновой кислоты, кодирующей TCR или его антигенсвязывающий фрагмент.
204. Сконструированная клетка по любому из вариантов осуществления 174-186 и 188-203, где полистронный элемент(элементы) расположен между последовательностью нуклеиновой кислоты, кодирующей TCRα или его часть, и последовательностью нуклеиновой кислоты, кодирующей TCRβ или его часть.
205. Сконструированная клетка по любому из вариантов осуществления 174-186 и 188-204, где один или более полицистронных элементов представляют собой или содержат последовательность пропуска рибосомы, необязательно где последовательность пропуска рибосомы представляет собой элемент T2A, P2A, E2A или F2A.
206. Сконструированная клетка по любому из вариантов осуществления 174-186 и 188-205, где трансгенная последовательность или последовательность нуклеиновой кислоты, кодирующая TCR или его антигенсвязывающий фрагмент, содержит один или более гетерологичных или регуляторных контрольных элементов, функционально связанных с контролем экспрессии TCR, когда он экспрессируется сконструированной клеткой, в которую он был введен.
207. Сконструированная клетка по варианту осуществления 206, где один или более гетерологичных регуляторных или контрольных элементов содержат промотор, энхансер, интрон, сигнал полиаденилирования, консенсусную последовательность Козака, акцепторную последовательность сплайсинга и/или донорную последовательность сплайсинга.
208. Сконструированная клетка по варианту осуществления 206 или 207, где гетерологичный регуляторный или контрольный элемент содержит гетерологичный промотор, необязательно промотор фактора элонгации 1 альфа (EF1α) человека или промотор MND или его вариант.
209. Сконструированная клетка по любому из вариантов осуществления 139-208, где TCR или его антигенсвязывающий фрагмент или его часть являются гетерологичными по отношению к клетке.
210. Сконструированная клетка по любому из вариантов осуществления 139-209, где сконструированная клетка представляет собой клеточную линию.
211. Сконструированная клетка по любому из вариантов осуществления 139-210, где сконструированная клетка представляет собой первичную клетку, полученную от пациента.
212. Сконструированная клетка по варианту осуществления 211, где пациентом является млекопитающее.
213. Сконструированная клетка по варианту осуществления 211 или 212, где пациентом является человек.
214. Сконструированная клетка по любому из вариантов осуществления 139-213, где сконструированная клетка представляет собой Т-клетку.
215. Сконструированная клетка по варианту осуществления 214, где Т-клетка представляет собой CD8+ Т-клетку.
216. Сконструированная клетка по варианту осуществления 214, где Т-клетка представляет собой CD4+ Т-клетку.
217. Способ получения клетки по любому из вариантов осуществления 139-216, включающий введение вектора по любому из вариантов осуществления 130-138 в клетку in vitro или ex vivo.
218. Способ получения клетки, включающий введение молекулы нуклеиновой кислоты, кодирующей TCR или его антигенсвязывающий фрагмент, по любому из вариантов осуществления 1-39 и 47-85, молекулы нуклеиновой кислоты по любому из вариантов осуществления 40-46 и 86-94, полинуклеотида по любому из вариантов осуществления 95-129 или вектора по любому из вариантов осуществления 130-138 в клетку in vitro или ex vivo.
219. Способ по варианту осуществления 217 или 218, где вектор представляет собой вирусный вектор, а введение осуществляют трансдукцией.
220. Способ по любому из вариантов осуществления 217-219, дополнительно включающий введение в клетку одного или более агентов, где каждый из одного или более агентов независимо способен индуцировать генетическое нарушение гена константной области Т-клеточного рецептора альфа (TRAC) и/или гена константной области Т-клеточного рецептора бета (TRBC).
221. Способ получения сконструированной клетки, включающий:
(i) введение молекулы нуклеиновой кислоты, кодирующей TCR или его антигенсвязывающий фрагмент по любому из вариантов осуществления 1-39 и 47-85, молекулы нуклеиновой кислоты по любому из вариантов осуществления 40-46 и 86-94, полинуклеотида по любому из вариантов осуществления 95-129 или вектора по любому из вариантов осуществления 130-138 в клетку; и
(ii) введение в клетку одного или более агентов, где каждый из одного или более агентов независимо способен индуцировать генетическое нарушение гена константной области Т-клеточного рецептора альфа (TRAC) и/или гена константной области Т-клеточного рецептора бета (TRBC).
222. Способ по варианту осуществления 221, где ген TRBC представляет собой один или оба из гена константной области 1 Т-клеточного рецептора бета (TRBC1) или гена константной области 2 Т-клеточного рецептора бета (TRBC2).
223. Способ по любому из вариантов осуществления 221-222, где один или более агентов, способных индуцировать генетическое нарушение, включают ДНК-связывающий белок или ДНК-связывающую нуклеиновую кислоту, которые специфически связываются с сайтом-мишенью или гибридизуются с ним.
224. Способ по варианту осуществления 223, где один или более агентов, способных индуцировать генетическое нарушение, включают (a) слитый белок, содержащий ДНК-нацеленный белок и нуклеазу, или (b) РНК-направляемую нуклеазу.
225. Способ по варианту осуществления 224, где ДНК-нацеленный белок или РНК-направляемая нуклеаза содержат белок цинкового пальца (ZFP), белок TAL или нуклеазу Cas, ассоциированную с нуклеиновой кислотой CRISPR (короткие палиндромные повторы, регулярно расположенные группами), специфичную для сайта-мишени в гене TRAC и/или TRBC.
226. Способ по варианту осуществления 225, где один или более агентов включают нуклеазу цинкового пальца (ZFN), эффекторную нуклеазу TAL (TALEN) или комбинацию CRISPR-Cas9, которая специфически связывается, распознает или гибридизуется с сайтом-мишенью.
227. Способ по варианту осуществления 225 или 226, где каждый из одного или более агентов содержит гид-РНК (гРНК), имеющую нацеливающий домен, который комплементарен по меньшей мере одному сайту-мишени.
228. Способ по вариантам осуществления 217-227, где ген TRBC представляет собой один или оба из гена константной области 1 Т-клеточного рецептора бета (TRBC1) или гена константной области 2 Т-клеточного рецептора бета (TRBC2).
229. Способ по любому из вариантов осуществления 221-228, где на генетическое нарушение воздействует один или более агентов, которые включают (а) по меньшей мере одну гРНК, имеющую нацеливающий домен, который комплементарен домену-мишени гена TRAC и/или TRBC, или (b) по меньшей мере одну нуклеиновую кислоту, кодирующую по меньшей мере одну гРНК.
230. Способ по варианту осуществления 229, где один или более агентов вводят в виде рибонуклеопротеинового комплекса (RNP), содержащего гРНК и белок Cas9.
231. Способ по варианту осуществления 230, где RNP вводят посредством электропорации, генной пушки, трансфекции фосфатом кальция, сжатия или сдавливания клеток.
232. Способ по варианту осуществления 230 или 231, где RNP вводят посредством электропорации.
233. Способ по любому из вариантов осуществления 227-232, где один или более агентов вводят в виде одного или более полинуклеотидов, кодирующих гРНК и/или белок Cas9.
234. Способ по любому из вариантов осуществления 220-233, где один или более агентов содержат по меньшей мере один комплекс молекулы Cas9 и по меньшей мере одну гРНК, имеющую нацеливающий домен, который комплементарен домену-мишени гена TRAC и/или гена TRBC.
235. Способ по любому из вариантов осуществления 220-234, где один или более агентов содержат, по меньшей мере, один комплекс из молекулы Cas9 и гРНК, имеющей нацеливающий домен, который представляет собой один или оба из (1) комплементарного домену-мишени гена TRAC, причем указанный нацеливающий домен содержит последовательность, выбранную из любой из SEQ ID NO: 1048 и 1229-1258; и (2) комплементарного домену-мишени гена TRBC, необязательно одного или обоих из генов TRBC1 и TRBC2, причем указанный домен-мишень содержит последовательность, выбранную из любой из SEQ ID NO: 1053 и 1259-1315.
236. Способ по любому из вариантов осуществления 229-235, где нацеливающий домен содержит последовательность, комплементарную домену-мишени гена TRAC, и нацеливающий домен содержит последовательность GAGAAUCAAAAUCGGUGAAU (SEQ ID NO: 1048).
237. Способ по любому из вариантов осуществления 229-236, где нацеливающий домен содержит последовательность, комплементарную домену-мишени гена TRBC, и нацеливающий домен содержит последовательность GGCCUCGGCGCUGACGAUCU (SEQ ID NO: 1053).
238. Способ по любому из вариантов осуществления 229-237, где гид-РНК дополнительно содержит первый домен комплементарности, второй домен комплементарности, который комплементарен первому домену комплементарности, проксимальный домен и, необязательно, хвостовой домен.
239. Способ по варианту осуществления 238, где первый домен комплементарности и второй домен комплементарности соединены связывающим доменом.
240. Способ по любому из вариантов осуществления 239, где гид-РНК содержит 3'-поли-А-хвост и 5'-Cap-аналог (Anti-Reverse Cap Analog) (ARCA).
241. Способ по любому из вариантов осуществления 226-240, где молекула Cas9 представляет собой ферментативно активную Cas9.
242. Способ по любому из вариантов осуществления 226-241, где молекула Cas9 представляет собой молекулу Cas9 S. aureus.
243. Способ по любому из вариантов осуществления 226-241, где молекула Cas9 представляет собой Cas9 S. pyogenes.
244. Способ по любому из вариантов осуществления 220-243, отличающийся тем, что, по меньшей мере или более чем 35%, 40%, 45%, 50%, 55%, 60%, 65%, 70%, 75%, 80%, 90% или 95% клеток во множестве сконструированных клеток содержат генетическое нарушение в гене TRAC и/или гене TRBC.
245. Способ по любому из вариантов осуществления 220-244, где по меньшей мере или более чем 90%, 95%, 96%, 97% или 98% клеток во множестве сконструированных клеток содержат генетическое нарушение в гене TRAC и/или гене TRBC.
246. Способ по любому из вариантов осуществления 217-245, где по меньшей мере или более чем 5%, 10%, 15%, 20%, 25%, 30%, 40%, 50% или более клеток во множестве сконструированных клеток экспрессируют введенный TCR или его антигенсвязывающий фрагмент и/или проявляют антигенное связывание с белком HPV, необязательно HPV E6 или HPV E7.
247. Способ по любому из вариантов осуществления 217-245, где по меньшей мере или более чем 5%, 10%, 15%, 20%, 25%, 30%, 40%, 50% или более клеток во множестве сконструированных клеток экспрессируют введенный TCR или его антигенсвязывающий фрагмент и/или проявляют антигенное связывание с белком HPV, необязательно HPV E6 или HPV E7.
248. Способ по любому из вариантов осуществления 217-247, где по меньшей мере или более чем 5%, 10%, 15%, 20%, 25%, 30%, 40%, 50% или более клеток во множестве сконструированных клеток экспрессируют введенный TCR или его антигенсвязывающий фрагмент и/или проявляют антигенное связывание с белком HPV, необязательно HPV E6 или HPV E7.
249. Способ по любому из вариантов осуществления 220-248, где один или более агентов и молекулу нуклеиновой кислоты, полинуклеотид или вектор вводят одновременно или последовательно в любом порядке.
250. Способ по любому из вариантов осуществления 220-251, где молекулу нуклеиновой кислоты, полинуклеотид или вектор вводят после введения одного или более агентов.
251. Способ по варианту осуществления 250, где молекулу нуклеиновой кислоты, полинуклеотид или вектор вводят сразу после или в течение примерно 30 секунд, 1 минуты, 2 минут, 3 минут, 4 минут, 5 минут, 6 минут, 6 минут, 8 минут, 9 минут, 10 минут, 15 минут, 20 минут, 30 минут, 40 минут, 50 минут, 60 минут, 90 минут, 2 часов, 3 часов или 4 часов после введения агента.
252. Композиция, содержащая сконструированные клетки по любому из вариантов осуществления 139-216.
253. Композиция, содержащая сконструированные клетки, полученные с применением способа по любому из вариантов осуществления 217-251.
254. Композиция по варианту осуществления 252 или 253, отличающаяся тем, что:
по меньшей мере 70%, 75%, 80%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% сконструированных клеток в композиции содержат генетическое разрушение эндогенного гена константной области Т-клеточного рецептора альфа (TRAC) и/или гена константной области Т-клеточного рецептора бета (TRBC); и/или
по меньшей мере 80%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% сконструированных клеток в композиции не экспрессируют или не экспрессируют на детектируемом уровне генный продукт эндогенного гена TRAC или TRBC.
255. Композиция, содержащая множество сконструированных клеток, причем каждая содержит TCR или его антигенсвязывающий фрагмент, где:
(1) по меньшей мере 70%, 75%, 80%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% сконструированных клеток в композиция содержит генетическое нарушение эндогенного гена константной области T-клеточного рецептора альфа (TRAC) и/или гена константной области T-клеточного рецептора бета (TRBC), и/или где по меньшей мере 80%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% сконструированных клеток в композиции не экспрессируют или не экспрессируют на детектируемом уровне генный продукт эндогенного гена TRAC или TRBC; и
(2) TCR или его антигенсвязывающий фрагмент содержат:
(а) вариабельную область альфа (Vα), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 117, 119 или 295, или аминокислотную последовательность, которая имеет с ними по меньшей мере 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, или 99% идентичности последовательности, и вариабельную область бета (Vβ), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 118, 120 или 296, или аминокислотную последовательность, которая имеет с ними по меньшей мере 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, или 99% идентичности последовательности; и/или
(b) область Vα, содержащую область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 153, 159 или 301, или CDR3, содержащуюся в аминокислотной последовательности, представленной в любой из SEQ ID NO: 117, 119 или 295, и область Vβ, содержащую область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 156 или 160, или CDR3, содержащуюся в аминокислотной последовательности, представленной в любой из SEQ ID NO: 118, 120 или 296.
256. Композиция по варианту осуществления 255, отличающаяся тем, что:
область Vα дополнительно содержит область 1, определяющую комплементарность, (CDR-1), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 151 или 157; и/или область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 152 или 158; и/или
область Vβ дополнительно содержит область 1, определяющую комплементарность, (CDR-1), содержащую аминокислотную последовательность, представленную в SEQ ID NO: 154; и/или область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность, представленную в SEQ ID NO: 155.
257. Композиция по варианту осуществления 255 или 256, где область Vα и область Vβ содержат:
CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 151, 152 и 153, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 156, соответственно;
CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 157, 158 и 159, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 160, соответственно; или
CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 151, 152 и 301, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 154, 155 и 156, соответственно.
258. Композиция по любому из вариантов осуществления 255-257, отличающаяся тем, что:
область Vα содержит область 1, определяющую комплементарность, (CDR-1), CDR-2 и CDR-3, соответственно, содержащие аминокислотные последовательности CDR-1, CDR-2 и CDR-3, содержащиеся в аминокислотной последовательности области Vα, представленной в любой из SEQ ID NO: 117, 119 или 295; и/или
область Vβ содержит область 1, определяющую комплементарность, (CDR-1), CDR-2 и CDR-3, соответственно, содержащие аминокислотные последовательности CDR-1, CDR-2 и CDR-3, содержащиеся в аминокислотной последовательности области Vβ, представленной в любой из SEQ ID NO: 118, 120 или 296.
259. Композиция по любому из вариантов осуществления 255-258:
области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 117 и либо 118, либо 296, соответственно;
области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 119 и 120, соответственно; или
области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 295 и либо 118, либо 296, соответственно.
260. Композиция по любому из вариантов осуществления 255-259, где TCR или его антигенсвязывающий фрагмент связывается с пептидным эпитопом вируса папилломы человека (HPV) 16 Е7 или распознает его в контексте молекулы МНС, причем пептидный эпитоп представляет собой или содержит Е7 (11-19) YMLDLQPET (SEQ ID NO: 236).
261. Композиция, содержащая множество сконструированных клеток, причем каждая содержит TCR или его антигенсвязывающий фрагмент, где:
(1) по меньшей мере 70%, 75%, 80%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% сконструированных клеток в композиция содержит генетическое нарушение эндогенного гена константной области T-клеточного рецептора альфа (TRAC) и/или гена константной области T-клеточного рецептора бета (TRBC), и/или где по меньшей мере 80%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% сконструированных клеток в композиции не экспрессируют или не экспрессируют на детектируемом уровне генный продукт эндогенного гена TRAC или TRBC; и
(2) TCR или его антигенсвязывающий фрагмент или рекомбинантный TCR или его антигенсвязывающий фрагмент содержат:
(a) вариабельную область альфа (Vα), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 111, 113, 115, 121, 123, 125, 297 или 299, или аминокислотную последовательность, которая имеет с ними по меньшей мере 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% идентичности последовательности; и/или область Vβ, содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: SEQ ID NO: 112, 114, 116, 122, 124, 126, 298 или 300, или аминокислотную последовательность, которая имеет с ними по меньшей мере 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% идентичности последовательности; и/или
(b) область Vα, содержащую область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 138, 144, 147, 163, 167, 173, 304 или 308, или CDR3, содержащуюся в аминокислотной последовательности, представленной в любой из SEQ ID NO: 111, 113, 115, 121, 123, 125, 297 или 299, и область Vβ, содержащую область 3, определяющую комплементарность, (CDR-3), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 141, 146, 150, 164, 170, 174, 305 или 309, или CDR3, содержащуюся в аминокислотной последовательности, представленной в любой из SEQ ID NO: 112, 114, 116, 122, 124, 126, 298 или 300.
262. Композиция по варианту осуществления 261, отличающаяся тем, что:
область Vα дополнительно содержит область 1, определяющую комплементарность, (CDR-1), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 136, 142, 161, 165, 171, 302 или 306, или CDR-1, содержащуюся в аминокислотной последовательности, представленной в любой из SEQ ID NO: 111, 113, 115, 121, 123, 125, 297 или 299; и/или область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 137, 143, 162, 166, 172, 303 или 307, или CDR-2, содержащуюся в аминокислотной последовательности, представленной в любой из SEQ ID NO: 111, 113, 115, 121, 123, 125, 297 или 299; и/или
область Vβ дополнительно содержит область 1, определяющую комплементарность, (CDR-1), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 139, 145, 148, 168, или CDR-1, содержащуюся в аминокислотной последовательности, представленную в любой из SEQ ID NO: 112, 114, 116, 122, 124, 126, 298 или 300; и/или область 2, определяющую комплементарность, (CDR-2), содержащую аминокислотную последовательность, представленную в любой из SEQ ID NO: 140, 149 или 169, или CDR-2, содержащуюся в аминокислотной последовательности, представленной в любой из SEQ ID NO: 112, 114, 116, 122, 124, 126, 298 или 300.
263. Композиция по варианту осуществления 261 или 262, отличающаяся тем, что:
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 136, 137 и 138, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 139, 140 и 141, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 142, 143 и 144, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 145, 140 и 146, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 136, 137 и 147, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 150, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 161, 162 и 163, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 164, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 165, 166 и 167, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 168, 169 и 170, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 171, 172 и 173, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 174, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 302, 303 и 304, соответственно, а область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 139, 140 и 305, соответственно;
область Vα содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 306, 307 и 308, соответственно, и область Vβ содержит CDR-1, CDR-2 и CDR-3, содержащие аминокислотные последовательности SEQ ID NO: 148, 149 и 309, соответственно.
264. Композиция по любому из вариантов осуществления 261-263, отличающаяся тем, что:
область Vα содержит область 1, определяющую комплементарность, (CDR-1), CDR-2 и CDR-3, соответственно, содержащие аминокислотные последовательности CDR-1, CDR-2 и CDR-3, содержащиеся в аминокислотной последовательности области Vα, представленной в любой из SEQ ID NO: 111, 113, 115, 121, 123, 125, 297 или 299; и/или
область Vβ содержит область 1, определяющую комплементарность, (CDR-1), CDR-2 и CDR-3, соответственно содержащие аминокислотные последовательности CDR-1, CDR-2 и CDR-3, содержащиеся в аминокислотной последовательности области Vβ, представленной в любой из SEQ ID NO: 112, 114, 116, 122, 124, 126, 298 или 300.
265. Композиция по любому из вариантов осуществления 261-264: области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 111 и 112, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 113 и 114, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 115 и 116, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 121 и 122, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 123 и 124, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 125 и 126, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 297 и 298, соответственно, области Vα и Vβ содержат аминокислотные последовательности SEQ ID NO: 299 и 300, соответственно.
266. Композиция по любому из вариантов осуществления 261-265, где TCR или его антигенсвязывающий фрагмент связывается или распознает пептидный эпитоп вируса папилломы человека (HPV) 16 Е6 в контексте молекулы МНС, причем пептидный эпитоп представляет собой или содержит Е6 (29-38) TIHDIILECV (SEQ ID NO: 233).
267. Композиция по любому из вариантов осуществления 255-266, где альфа-цепь дополнительно содержит константную область альфа (Cα), и/или бета-цепь дополнительно содержит константную область бета (Cβ).
268. Композиция по любому из вариантов осуществления 252-267, где по меньшей мере или более чем 90%, 95%, 96%, 97% или 98% клеток в композиции содержат генетическое нарушение эндогенного гена TRAC и/или гена TRBC и/или не экспрессируют или не экспрессируют на детектируемом уровне генный продукт эндогенного гена TRAC или TRBC.
269. Композиция по любому из вариантов осуществления 252-268, где по меньшей мере или более чем 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97% или 98% клеток в композиции i) экспрессируют сконструированный или рекомбинантный TCR или его антигенсвязывающий фрагмент и (ii) содержат генетическое нарушение эндогенного гена TRAC и/или гена TRBC и/или не экспрессируют или не экспрессируют на детектируемом уровне генный продукт эндогенного гена TRAC или TRBC.
270. Композиция по любому из вариантов осуществления 254-259, где ген TRBC представляет собой один или оба из гена константной области 1 Т-клеточного рецептора бета (TRBC1) или гена константной области 2 Т-клеточного рецептора бета (TRBC2).
271. Композиция по любому из вариантов осуществления 254-260, где генный продукт представляет собой мРНК или белок, кодируемый геном TRAC или TRBC.
272. Композиция по любому из вариантов осуществления 252-271, где сконструированные клетки содержат CD4+ и/или CD8+ Т-клетки.
273. Композиция по любому из вариантов осуществления 252-272, где сконструированные клетки содержат CD4+ и CD8+ Т-клетки.
274. Композиция, содержащая сконструированную CD8+ клетку по варианту осуществления 215 и сконструированную CD4+ клетку по варианту осуществления 216.
275. Композиция по любому из вариантов осуществления 252-274, где TCR или его антигенсвязывающий фрагмент связываются или распознают пептидный эпитоп HPV 16 в контексте молекулы МНС, что является по меньшей мере частично CD8-независимым.
276. Композиция по любому из вариантов осуществления 272-275, где CD8+ клетки и CD4+ клетки сконструированы с одним и тем же TCR или его антигенсвязывающим фрагментом, и/или каждая из них сконструирована с TCR или его антигенсвязывающим фрагментом, которые связываются или распознают тот же пептидный эпитоп HPV 16 в контексте молекулы МНС.
277. Композиция по любому из вариантов осуществления 254-276, отличающаяся тем, что:
генетическое нарушение включает мутацию или делецию в области гена TRAC или TRBC, которая находится в кодирующей области, необязательно в ранней кодирующей области гена, находится в пределах экзона 1 гена, находится в кодирующей области в пределах 500, 400 300, 200, 100 или 50 пар оснований от старт-кодона гена, находится в последовательности сайта-мишени, которая комплементарна нацеливающему сайту нацеливающего домена гРНК, имеющего последовательность, выбранную из любой из SEQ ID NO: 1053 и 1259-1315, и/или с которым специфически гибридизуется нацеливающий домен, имеющий последовательность, выбранную из SEQ ID NO: 1053 и 1259-1315, и/или находится в последовательности сайта-мишени, которая комплементарна нацеливающему сайту нацеливающего домена гРНК, имеющего последовательность, выбранную из любой из SEQ ID NO: 1048 и 1229-1258, и/или с которым специфически гибридизуется нацеливающий домен, имеющий последовательность, выбранную из SEQ ID NO: 1048 и 1229-1258.
278. Композиция по пп.274-277, где по меньшей мере одна из сконструированных клеток представляет собой клетку по любому из вариантов осуществления 139-216.
279. Композиция по любому из вариантов осуществления 252-278, где на генетическое нарушение воздействует один или более агентов, которые содержат (a) по меньшей мере одну гРНК, имеющую нацеливающий домен, который комплементарен домену-мишени гена TRAC и/или гена TRBC, или (b) по меньшей мере одну нуклеиновую кислоту, кодирующую по меньшей мере одну гРНК.
280. Композиция по варианту осуществления 279, где один или более агентов вводят в виде рибонуклеопротеинового (RNP) комплекса, содержащего гРНК и белок Cas9.
281. Композиция по варианту осуществления 280, где RNP вводят посредством электропорации, генной пушки, трансфекции фосфатом кальци, сжатия или сдавливания клеток.
282. Композиция по варианту осуществления 280 или п. 281, где RNP вводят электропорацией.
283. Композиция по любому из вариантов осуществления 279-282, где один или более агентов вводят в виде одного или более полинуклеотидов, кодирующих гРНК и/или белок Cas9.
284. Композиция по любому из вариантов осуществления 279-283, где один или более агентов содержат по меньшей мере один комплекс из молекулы Cas9 и по меньшей мере одной гРНК, имеющей нацеливающий домен, который комплементарен домену-мишени гена TRAC и/или гена TRBC.
285. Композиция по любому из вариантов осуществления 279-284, где один или более агентов содержат по меньшей мере один комплекс из молекулы Cas9 и гРНК, имеющей нацеливающий домен, который представляет собой один или оба из следующего: (1) комплементарен домену-мишени гена TRAC, причем указанный нацеливающий домен содержит последовательность, выбранную из любой из SEQ ID NO: 1048 и 1229-1258; и (2) комплементарен домену-мишени гена TRBC, необязательно одного или обоих из генов TRBC1 и TRBC2, причем указанный нацеливающий домен содержит последовательность, выбранную из любой из SEQ ID NO: 1053 и 1259-1315.
286. Композиция по любому из вариантов осуществления 279-285, где нацеливающий домен содержит последовательность, комплементарную домену-мишени гена TRAC, и нацеливающий домен содержит последовательность GAGAAUCAAAAUCGGUGAAU (SEQ ID NO: 1048).
287. Композиция по любому из вариантов осуществления 279-286, где нацеливающий домен содержит последовательность, комплементарную домену-мишени гена TRBC, и нацеливающий домен содержит последовательность GGCCUCGGCGCUGACGAUCU (SEQ ID NO: 1053).
288. Композиция по любому из вариантов осуществления 279-287, где гид-РНК дополнительно содержит первый домен комплементарности, второй домен комплементарности, который комплементарен первому домену комплементарности, проксимальный домен и, необязательно, хвостовой домен.
289. Композиция по варианту осуществления 288, где первый домен комплементарности и второй домен комплементарности соединены с помощью связывающего домена.
290. Композиция по любому из вариантов осуществления 289, где гид-РНК содержит 3'-поли-А-хвост и 5'-Cap-аналог (Anti-Reverse Cap Analog) (ARCA).
291. Композиция по любому из вариантов осуществления 279-290, где молекула Cas9 представляет собой ферментативно активную Cas9.
292. Композиция по любому из вариантов осуществления 279-291, где молекула Cas9 представляет собой молекулу Cas9 S. aureus.
293. Композиция по любому из вариантов осуществления 279-291, где молекула Cas9 представляет собой Cas9 S. pyogenes.
294. Композиция по любому из вариантов осуществления 252-293, дополнительно содержащая фармацевтически приемлемое вспомогательное вещество.
295. Способ лечения, включающий введение сконструированной клетки по любому из вариантов осуществления 139-216 пациенту, имеющему заболевание или расстройство, ассоциированное с HPV.
296. Способ лечения, включающий введение композиции по любому из вариантов осуществления 252-294 пациенту, имеющему заболевание или расстройство, ассоциированное с HPV.
297. Способ по варианту осуществления 295 или 296, где заболевание или расстройство ассоциировано с HPV16.
298. Способ по любому из вариантов осуществления 295-297, где заболевание или расстройство представляет собой онкологическое заболевание.
299. Способ по любому из вариантов осуществления 295-298, где пациентом является человек.
300. Композиция по любому из вариантов осуществления 252-294 для применения при лечении заболевания или расстройства, ассоциированного с HPV.
301. Применение композиции по любому из вариантов осуществления 252-294 для изготовления лекарственного средства для лечения заболевания или расстройства, ассоциированного с HPV.
302. Композиция по варианту осуществления 300 или применение по варианту осуществления 301, где заболевание или расстройство ассоциировано с HPV16.
303. Композиция или применение по любому из вариантов осуществления 300-302, где заболевание или расстройство представляет собой онкологическое заболевание.
304. Композиция или применение по любому из вариантов осуществления 300-303, где пациентом является человек.
X. Примеры
[0993] Следующие примеры включены только в иллюстративных целях и не предназначены для ограничения объема изобретения.
Пример 1. Скрининг и отбор Т-клеточных рецепторов, специфических для эпитопов E6 и E7 HPV-16, от здоровых доноров.
[0994] Пример процесса аутологичного скрининга с использованием аутологичных дендритных и Т-клеток, как в целом описано Ho et al., J. Immunol. Methods, 310:1-2, 40-52, с указанными модификациями, выполняли для генерирования антиген-специфических Т-клеток, которые специфически связывались с пептидными эпитопами белков E6 и E7 вируса папилломы человека 16 (HPV16), презентированных на молекулах MHC-I. Были получены клональные Т-клеточные линии, и их последовательности TCR, клонированные этим способом, были клонированы.
A. Генерация и клонирование человеческих HPV-специфических Т-клеток и TCR
[0995] Вкратце, дендритные клетки были получены из прикрепленных фракций образцов мононуклеарных клеток периферической крови (PBMC), полученных от здоровых доноров HLA-A02: 01 человека, путем культивирования в течение двух дней в присутствии GM-CSF и IL-4 с последующим началом инкубации в день 3 в присутствии провоспалительных цитокинов для продуцирования зрелых дендритных клеток. На 4-й день полученные зрелые дендритные клетки собирали, промывали и стимулировали пептидами, происходящими из Е6 или Е7 HPV-16, такими как некоторые из указанных в Таблице 23, включая пептидные эпитопы Е6 (29-38), Е7 (11-19) и E7 (86-93).
[0996] На 5-ый день аутологичные CD8+ Т-клетки от здоровых людей-доноров инкубировали с дендритными клетками, стимулированными пептидом.
[0997] На 8-ой день IFNγ в культурах измеряли в качестве индикатора для культур, содержащих антигенспецифические Т-клетки. Клетки из реактивных совместных культур отбирали и повторно стимулировали два или три раза с помощью стимулированных пептидом дендритных клеток для обогащения специфическими Т-клетками. После повторных стимуляций популяции клеток, окрашивающихся положительно в отношении нагруженных пептидом аутологичных тетрамеров МНС, были идентифицированы проточной цитометрией. Клональные линии были получены путем сортировки клеток и/или клонирования с использованием лимитирующего разведения, по существу, как описано Ho et al. 2006.
[0998] Клоны культивировали со стимулированными пептидом T2-клетками (клетками с дефицитом по белку-переносчику, ассоциированному с транспортом антигена (TAP), но экспрессирующими MHC-I и, таким образом, способными презентировать нагруженные пептиды на клетках), стимулированными соответствующим пептидом, например, Е6 (29-38), Е7 (11-19) или Е7 (86-93). Уровень IFNγ в культурах по сравнению с уровнями, полученными в результате совместного культивирования с клетками, нагруженными пептидом, не являющимся производным HPV (отрицательный контроль), измеряли как показатель специфичности Т-клеток к пептиду-МНС и функциональной активности. Окрашивание на основе проточной цитометрии использовали для оценки способности клональных клеточных линий связываться пептид-специфическим образом с мечеными пептид-MHC (HLA-A02: 01) тетрамерами (либо HLA-A2/E6 (29-38).), HLA-A2/E7 (11-19) или HLA-A2/E7 (86-93)); тетрамеры, содержащие нерелевантный пептид, служили отрицательным контролем).
[1000] В Таблице 24 перечислены идентификаторы последовательности, соответствующие альфа- и бета-цепям TCR, экспрессируемым клональными линиями Т-клеток, полученными в результате этого процесса.
[1001] Способность клональных линий лизировать клетки-мишени антигенспецифическим способом оценивали с использованием стимулированных пептидом Т2-клеток и/или клеток антиген-экспрессирующей линии опухолевых клеток.
[1002] В одном из примеров анализа моноклональные клеточные линии, экспрессирующие TCR, инкубировали с клетками-мишенями CaSki (ATCC No. CRL-1550, содержащие приблизительно 600 копий интегрированного HPV16) при различных соотношениях эффектор:мишень (E:T). Литическую активность оценивали, измеряя каспазу в клетках-мишенях и оценивая процент таких клеток, которые были положительными в отношении каспазы в различные моменты времени после начала инкубации с Т-клетками в течение 50 часов. Отрицательные контроли включали инкубацию Т-клеток с клетками SiHa (ATCC No. HTB-35, по существу, отрицательные в отношении эндогенного антигена-мишени, содержащие не более чем приблизительно одна или две копии интегрированного генома HPV16) и клетками Caski, не инкубированными с клонами Т-клеток. Результаты для двух иллюстративных клональных Т-клеточных линий показаны на ФИГ. 1. Как показано, наблюдали, что моноклональные Т-клеточные линии проявляют литическую активность против клеток, презентирующих рассматриваемый пептид, полученный из HPV16, в контексте HLA-A02: 01. Был создан ряд CD8+ клонов, и подтверждено, что они проявляют антиген-специфическое связывание и функциональность этим способом.
[1003] Способность Т-клеток клональных линий специфически связываться с пептидными эпитопами независимо от корецептора CD8 оценивали с использованием мутантного тетрамера MHC класса I, содержащего мутацию D227K в своем сайте связывания CD8, что делает его неспособным рекрутировать корецептор CD8 на Т-клетки. Смотри Kerry et al. J Immunol (2003) 171:4493-4503; Kerry et al. Immunology (2005) 114: 44-52. В Таблице 24 приведены иллюстративные TCR, экспрессируемые иллюстративными клональными клеточными линиями, сгенерированными этим способом. В этом исследовании наблюдали, что каждая из этих клеточных линий связывала указанный комплекс пептид-МНС антигенспецифическим образом, как показано с помощью окрашивания тетрамером по сравнению с контролем. Кроме того, наблюдали, что указанные клональные линии специфически связывают соответствующий пептид в контексте мутантных (не взаимодействующих с CD8) тетрамеров, что указывает на способность TCR, экспрессируемых этими клональными линиями, специфически связываться с распознаваемым антигеном независимо от CD8.
B. Клонирование TCR, экспрессируемых клональными клеточными линиями
[1004] Полинуклеотиды, имеющие последовательности, кодирующие полипептидные цепи TCR из клональных линий, полученных, как описано выше, амплифицировали из Т-клеточных линий и секвенировали с использованием 5'-быстрой амплификации концов кДНК (RACE). В Таблице 24 представлен идентификатор последовательности (SEQ ID NO) для нуклеотидной и аминокислотной последовательностей альфа- и бета-цепи, соответственно, для множества TCR, полученных этим способом. В Таблице 24 также перечислены SEQ ID NO, соответствующие иллюстративной полноразмерной кодированной аминокислотной последовательности, содержащей последовательности бета- и альфа-цепей каждого соответствующего TCR, разделенные последовательностью P2A пропуска рибосомы (линкер P2A, представленный в SEQ ID NO: 204, которая может кодироваться последовательностью нуклеотидов, представленной в любой из SEQ ID NO: 4, 5, 6, 207-210) (обозначена как «бета-P2A-альфа»). Нуклеотидная последовательность, кодирующая такую полноразмерную последовательность для каждого из ряда TCR, была вставлена в вектор для переноса в клетку-хозяина, такую как первичная клетка человека, например Т-клетка, как описано ниже. После трансляции нуклеотидной последовательности и саморасщепления последовательности P2A, разделяющей цепи TCR, рекомбинантные альфа- и бета-цепь TCR были экзогенно экспрессированы в клетках-хозяевах, таких как первичная Т-клетка, например первичная Т-клетка человека. В Таблице 24 также перечислены конкретные использования Vальфа и Vбета для каждого клонированного TCR.
Vальфа
Vбета
C. Оптимизация кодонов, модификация и лентивирусная экспрессия
[1005] Нуклеотидные последовательности, кодирующие TCR, полученные, как описано выше, были модифицированы путем оптимизации кодонов и/или мутации (мутаций) для ускорения образования ненативной дисульфидной связи на границе раздела между константными доменами TCR для увеличения спаривания и стабильности TCR. Ненативная дисульфидная связь стимулировалась путем модификации цепей TCR в остатке 48 в Cα-области с Thr до Cys и в остатке 57 Cβ-области с Ser до Cys (см. Kuball et al. (2007) Blood, 109: 2331-2338). Соответствующая SEQ ID NO для полученных модифицированных нуклеотидных последовательностей и соответствующие кодированные аминокислотные последовательности для модифицированной версии каждого TCR показаны в Таблице 25.
[1006] Для отдельных TCR, модифицированных, как описано выше, были сгенерированы конструкции, которые содержали модифицированные нуклеотидные последовательности, кодирующие бета-цепь и альфа-цепь, соответственно, клонированных TCR, разделенных последовательностью, кодирующей полипептид P2A, и были вставлены в лентивирусный вектор, который был использован для трансдукции Т-клеточных линий и первичных человеческих Т-клеток, используя стандартные методы, для экспрессии кодированных цепей TCR.
Пример 2. Экспрессия и связывание антигена иллюстративных TCR в клетках Jurkat.
[1007] Иллюстративные E6-специфические и E7-специфические T-клеточные рецепторы (TCR), полученные, как описано выше, оценивали на предмет поверхностной экспрессии на T-клетках и антиген-специфического связывания с или без взаимодействия CD8. В частности, клетки, полученные из линии человеческих T-клеток Jurkat, которые не экспрессировали эндогенный TCR на своих поверхностях (CD4+ клетки, полученные из Jurkat), с или без экзогенно экспрессируемого CD8, упоминаемые на ФИГ. 2А-2L, ФИГ. 3А-3D и ФИГ. 4A-4B, как CD8+ и CD4+, соответственно, были сконструированы для экспрессии модифицированной версии TCR. Для каждого TCR, оцененного в этом процессе, клетки, полученные из Jurkat, трансдуцировали лентивирусной векторной частицей, генерированной, как описано выше, кодирующей конкретную модифицированную версию TCR. Клетки (содержащие или не содержащие экзогенный CD8), не трансдуцированные TCR, использовали в качестве контролей. На 6-й день после трансдукции с последовательностью, кодирующей каждый TCR, экспрессию и функциональную активность TCR оценивали с помощью проточной цитометрии, после окрашивания мечеными тетрамерами в комплексе с соответствующим пептидом E6 или E7 (либо тетрамер HLA-A2/E6 (29-38), HLA-A2/E7 (11-19), либо HLA-A2/E7 (86-93). Эталонный TCR, способный связываться с HLA-A2/E6 (29-38), также оценивался в этом исследовании (описано в международной публикации PCT №. и WO 2015/009606). Эталонный TCR содержит константную область мыши.
[1008] Примеры результатов показаны на ФИГ. 2A-2L (связывание тетрамера с E6 (29-38)-нагрузкой), ФИГ. 3A-3D (связывание тетрамера с E7 (11-19) -нагрузкой) и ФИГ. 4A-4B (связывание тетрамера с E7 (86-93) -нагрузкой). Процент клеток в указанных квадрантах на графиках проточной цитометрии, показанных на ФИГ. 2A-2L, 3A-3D и 4A-4B также суммированы ниже в Таблице 26A (ФИГ.2A-2F), Таблице 26B (ФИГ.2G-2L), Таблице 26C (ФИГ.3A-3D) и Таблице 26D (ФИГ.4А-4В).
квадрант
квадрант
квадрант
Квадрант
квадрант
квадрант
квадрант
квадрант
[1009] Как показано, TCR, полученные этими способами, были клонированы, и было обнаружено, что они экспрессируются на поверхности Т-клеток и связываются с пептидом HPV в контексте тетрамеров MHC, в некоторых случаях независимо от корецептора CD8.
Пример 3. Функциональная оценка клеток, трансдуцированных Т-клеточными рецепторами, специфичными для эпитопов E6 и E7 HPV-16.
[1010] Первичные человеческие CD8+ T-клетки трансдуцировали лентивирусной векторной частицей, сгенерированной, как описано выше, кодирующей цепи модифицированных версий TCR, специфичных для E6 (29-38) в контексте HLA: A2: 01, включая примеры модифицированных версий TCR TCR 5, TCR 4, TCR 3, TCR 8, TCR 9, TCR 10 и TCR7. Такие трансдуцированные Т-клетки оценивали на функциональную активность, в том числе способность генерировать цитокины и проявлять литическую активность в ответ на клетки, экспрессирующие пептид: МНС. Иллюстративный E7 (11-19) -специфичный TCR использовали в качестве отрицательного контроля в этих исследованиях.
A. Продуцирование цитокинов
[1011] Для оценки прдуцирования цитокинов в ответ на антиген клетки инкубировали в течение 4 часов при соотношении E:T 10:1 с клетками T2, которые стимулировали в течение ночи с 10 мкМ пептида E6 (29-38) или, в качестве контроля, с 10 мкМ E7 (11-19) пептида. В качестве положительного контроля активность цитокинов также оценивали в культурах трансдуцированных Т-клеток, стимулированных либо форболи миристатацетатом (PMA) и брефелдином A (BFA), либо только BFA. Внутриклеточный IFNγ измеряли в культивируемых клетках методом проточной цитометрии. Процент CD8 и внутриклеточных IFNγ-положительных (% CD8+/IC IFNγ +) клеток определяли проточной цитометрией.
[1012] Результаты показаны в Таблице 27. Эти результаты подтвердили способность первичных человеческих Т-клеток, экспрессирующих E6 (29-38) -специфичные TCR, генерируемые этими методами, продуцировать цитокин в ответ на клетки-мишени антигенспецифическим образом.
B. Литическая активность
[1013] Литическую активность трансдуцированных первичных Т-клеток человека в отношении клеток, экспрессирующих HPV16, оценивали путем инкубации клеток CaSki (в присутствии или в отсутствие IFNγ) при соотношении E:T 10:1. Образцы, в которых клетки SiHa использовались в качестве клеток-мишеней при том же отношении E:T, служили отрицательным контролем. Литическую активность также оценивали в отношении клеток Т2, стимулированных пептидом Е6 (29-38). Способность Т-клеток антиген-специфически вызывать литическую активность оценивали путем измерения активной каспазы в клетках-мишенях через 4 часа после совместного культивирования.
Пример 4. Скрининг и отбор Т-клеточных рецепторов, специифических для эпитопов E6 и E7 HPV-16, от здоровых доноров.
[1014] Процесс скрининга с использованием аутологичных дендритных и Т-клеток проводили для генерирования антиген-специфических Т-клеточных рецепторов (TCR), которые специфически связываются с пептидом вируса папилломы человека 16 (HPV16), E6 (29-38) или E7 (11-19), презентированном на молекулах MHC- I, и выживают и/или обогащаются с течением времени после нескольких раундов антигенной стимуляции. Были получены клональные Т-клеточные линии и были определены последовательности отдельных парных альфа- и бета-цепей TCR и их содержание в различных популяциях на основе одной клетки с использованием высокопроизводительного секвенирования парного TCR.
A. Генерация и клонирование человеческих HPV-специфических Т-клеток и TCR
[1015] Вкратце, пептид-стимулированные антиген-презентирующие клетки были получены из РВМС от людей-доноров, по существу, как описано в Примере 1. В частности, пептид-стимулированные HLA: A02: 01APC были получены с пептидом E6 HPV 16 (29-38) (TIHDIILECV; SEQ ID NO: 233) или пептидом E7 (11-19) (YMLDLQPET; SEQ ID NO: 236). Аутологичные CD8+ Т-клетки от здоровых людей-доноров инкубировали в течение нескольких циклов с пептид-стимулированными клетками, и отбор проводили на основе связывания с нагруженными пептидом аутологичными тетрамерами МНС. Как правило, клетки подвергали в общей сложности трем циклам стимуляции в присутствии пептид-стимулированных клеток (с концентрацией пептида 1000 нг/мл, поддерживаемой в течение трех циклов). После второго и третьего раундов стимуляции клетки сортировали с помощью проточной цитометрии на популяции, положительные и отрицательные, соответственно, для связывания с тетрамерами пептид-МНС, содержащими соответствующий тетрамер. Клетки тетрамер-положительных и отрицательных популяций после каждого из второго и третьего раундов подвергали секвенированию TCR для одной клетки, чтобы оценить наличие и частоту отдельных TCR в разных популяциях, а также устойчивость клонов TCR в течение нескольких раундов антигенной стимуляции.
B. Определение последовательностей TCR и оценка TCR
[1016] Клеточные популяции из положительной и отрицательной фракций (то есть отсортированные методом проточной цитометрии на основе положительного и отрицательного окрашивания, соответственно, для связывания с E6 (29-38) пептид-нагруженным или E7 (11-19) пептид-нагруженным, MHC тетрамер, как определено методом проточной цитометрии) после раундов 2 и 3 стимуляции подвергались высокопроизводительному секвенированию из одной клетки для пар альфа- и бета-цепей TCR. Высокопроизводительное секвенирование TCR из одной клетки осуществляли, как обычно описано в опубликованных патентных заявках PCT, публикациях №№ WO2012/048340, WO2012/048341 и WO2016/044227. В методах секвенирования использовались капли из одной клетки, а также образцы и молекулярные штрих-коды, чтобы идентифицировать отдельные пары последовательностей альфа- и бета-цепей TCR на уровне одной клетки для каждой из большого числа (например, миллионов) отдельных клеток, присутствующих в одной исходной композиции, и оценить предстваленность каждой пары TCR в различных оцениваемых популяциях. Способность идентифицировать и количественно определять пары TCR на уровне отдельных клеток позволила оценить частоту каждой из различных пар TCR в каждой из отдельных положительных и отрицательных фракций, а также оценить обогащение и стабильность TCR в течение нескольких циклов антигенной стимуляции. Пары TCR, идентифицированные в этом анализе, были отобраны на основании их присутствия в пептидсвязывающих фракциях после раундов 2 и 3, с более высоким содержанием в положительных и отрицательных фракциях в каждом из этих раундов и обогащением с течением времени после нескольких раундов воздействия антигена.
[1017] В Таблицах 28 и 29 перечислены иллюстративные E6 (29-38)- и E7 (11-19) -специфичные TCR, выделенные согласно этому способу, соответственно, и идентификаторы последовательности (SEQ ID NO:) для нуклеотидной и аминокислотной последовательности альфа- и бета-цепи для каждого TCR. В Таблицах 28 и 29 также указан идентификатор последовательности (SEQ ID NO), соответствующий иллюстративной полноразмерной кодированной аминокислотной последовательности, содержащей последовательности бета и альфа-цепи каждого соответствующего TCR, разделенные последовательностью, кодирующей последовательность P2A пропуска рибосомы (P2A-линкер, представленный в SEQ ID NO: 204) (обозначен как «бета-P2A-альфа»). Нуклеотидная последовательность, кодирующая такую полноразмерную последовательность для каждого из ряда TCR, была вставлена в вектор для переноса в клетку-хозяина, такую как первичная клетка человека, например Т-клетка, как описано ниже. После трансляции нуклеотидной последовательности и саморасщепления последовательности P2A, разделяющей цепи TCR, рекомбинантные альфа- и бета-цепь TCR были экзогенно экспрессированы в клетках-хозяевах.
C. Оптимизация и модификация кодонов
[1018] Нуклеотидные последовательности, кодирующие TCR, полученные, как описано выше, были модифицированы путем оптимизации кодонов и/или мутации (мутаций) для стимулирования образования ненативной дисульфидной связи в интерфейсе между константными доменами TCR для увеличения спаривания и стабильности TCR. Ненативная дисульфидная связь стимулировалась путем модификации цепей TCR в остатке 48 в Cα-области с Thr до Cys и в остатке 57 Cβ-области с Ser до Cys (см. Kuball et al. (2007) Blood, 109: 2331-2338). Соответствующая SEQ ID NO для полученных модифицированных нуклеотидных последовательностей и соответствующие кодированные аминокислотные последовательности для модифицированной версии каждого TCR показаны в Таблице 30 (E6 (29-38) -специфичный TCR) и Таблице 31 (E7 (11-19) -специфичные TCR).
[1019] Для индивидуальных TCR, модифицированных, как описано выше, были получены конструкции, которые содержали модифицированные нуклеотидные последовательности, кодирующие бета-цепь и альфа-цепь, соответственно, клонированных TCR, разделенных последовательностью, кодирующей полипептид P2A, и вставленные в вектор, например, лентивирусный вектор, который использовали для экспрессии цепи TCR в Т-клеточных линиях и первичных Т-клетках человека с использованием стандартных методов.
Пример 5. Экспрессия и связывание антигена иллюстративных E6- и E7-специфических TCR.
[1020] Иллюстративные E6- и E7-специфические T-клеточные рецепторы (TCR), идентифицированные, как описано в Примере 4 выше, которые были оптимизированы по кодонам и модифицированы по цистеину, были экспрессированы в T-клетках и оценены на предмет поверхностной экспрессии и антиген-специфического связывания, с или без взаимодействия CD8, по существу, как описано в Примере 2 выше. В частности, CD4+ клетки, полученные из клеток Jurkat, которые не экспрессировали эндогенный TCR на своих поверхностях, которые были или не были модифицированы введением экзогенного CD8 (модификация, приводящая к получению CD4+/CD8+ клеток), смешивали в смеси 1:1 для трансфекции с плазмидной ДНК, кодирующей TCR, для оценки CD8-независимой активности связывания TCR. Для трансфекции смесь CD4+ и CD4+/CD8+ клеток временно трансфецировали плазмидами, кодирующими TCR, и через 48 часов после трансфекции клетки оценивали проточной цитометрией на предмет (1) связывания пептида-мишени в контексте молекулы МНС (HLA: A02: 01) путем окрашивания EH (29-38) пептид- или E7 (11-19) пептид-MHC тетрамерного реагента и/или (2) CD8+ независимого связывания мишени путем совместного окрашивания меченных тетрамером клеток с антителом против CD8. Клетки, которые были ложно трансфецированы (имитация), и клетки, экспрессирующие эталонный TCR, способный связываться с HLA-A2/E6 (29-38), также оценивали в этом исследовании.
[1021] Примеры результатов показаны на ФИГ. 5A-5F (связывание E6 (29-38) -нагруженного тетрамера) и ФИГ. 6A-6F (связывание E7 (11-19) -нагруженного тетрамера). Процент клеток в указанных квадрантах на графиках проточной цитометрии, показанных на ФИГ. 5A-5H и 6A-6H также суммирован ниже в Таблице 32 (графики проточной цитометрии, показывающие результаты окрашивания тетрамера E6 (29) и CD8+ для CD8+ клеток из композиций, трансфецированных TCR; ФИГ. 5A-5C), Таблица 33 (графики проточной цитометрии, показывающие результаты для E6 (29) -специфических TCR-трансфецированных клеточных композиций; ФИГ. 5D-5F) и Таблица 34 (графики проточной цитометрии, показывающие результаты для E7 (11) -специфичных TCR; ФИГ. 6A-6F). В частности, на ФИГ. 5А-5С представлены графики проточной цитометрии для окрашивания тетрамера и CD8 в CD8+ популяциях; На ФИГ. 5D-5F и 6A-6F показаны графики, отражающие окрашивание CD8+ и CD8-популяций.
квадрант
квадрант
квадрант
квадрант
квадрант
квадрант
квадрант
Квадрант
[1022] Как показано, иллюстративные оцениваемые TCR экспрессировались на поверхности Т-клеток и распознавали пептид HPV в контексте тетрамеров MHC. В некоторых случаях связывание не зависело от корецептора CD8, на что указывают тетрамер+клетки в CD8- популяции на ФИГ. 5D-5F (проценты указаны в Таблице 33) и на ФИГ. 6А-6F (проценты указаны в Таблице 34).
[1023] Результаты дополнительного исследования, оценивающего CD8-независимое связывание тетрамера клеток, экспрессирующих иллюстративные рекомбинантные E7-специфичные TCR, TCR37 и TCR49, которые были кодон-оптимизированы и модифицированы цистеином, показаны на ФИГ. 6G. Как показано, клетки, экспрессирующие иллюстративный рекомбинантный TCR, TCR49, как наблюдалось, демонстрировали CD8-зависимое связывание тетрамера (левая панель), и клетки, экспрессирующие иллюстративный рекомбинантный TCR, TCR37, как наблюдалось, демонстрировали некоторое CD8-независимое связывание тетрамера. Результаты согласуются с тем, что некоторые МНС класс I-рестриктированные рекомбинантные TCR могут функционировать в CD4+ клетках, демонстрируя независимое от CD8 связывание тетрамера.
Пример 6. Экспрессия и оценка иллюстративных рекомбинантных T-клеточных рецепторов (TCR) в первичных T-клетках.
[1024] Оценивали экспрессию и функцию иллюстративных рекомбинантных E7-специфических TCR в первичных Т-клетках человека.
[1025] Первичные человеческие CD4+ и CD8+ T-клетки трансдуцировали лентивирусными препаратами, кодирующими TCR 16, специфичными для E6 HPV 16 (29-38); и TCR 49, TCR 53 и TCR 37, каждый специфичный для E7 HPV 16 (11-19) (описанный выше в Примере 4 выше, и которые были оптимизированы по кодонам и модифицированы по цистеину). Приблизительно 5×106 первичных человеческих CD4+ и CD8+ T-клеток выделяли путем иммуноаффинного отбора из мононуклеарных клеток периферической крови человека (PBMC), полученных от здоровых доноров. Клетки стимулировали в течение 24 часов путем культивирования с реагентом анти-CD3/анти-CD28 в среде, содержащей человеческую сыворотку и цитокины, при 37°C перед лентивирусной трансдукцией. Стимулированные клетки трансдуцировали лентивирусным препаратом, кодирующим TCR 16, TCR 49, TCR 53 или TCR 37, или контролем ложной трансдукции (клетки обрабатывали в тех же условиях, что и для лентивирусной трансдукции, но без добавления лентивируса). Лентивирусные конструкции также содержали последовательности, кодирующие EGFRt в качестве суррогатного маркера для трансдукции и экспрессии, отделенные от последовательностей, кодирующих рекомбинантный TCR, последовательностью, кодирующей последовательность пропуска рибосомы T2A. После трансдукции клетки культивировали в среде, содержащей человеческую сыворотку и цитокины. На 13-й день после трансдукции клетки оценивали проточной цитометрией на предмет окрашивания антителом против CD3, антителом против CD8 и E6 HPV 16 (29-38)- или E7 HPV 16 (11-19)-пептид-МНС тетрамерным комплексом. Продуцирование интерферона-гамма (IFNγ) оценивали после инкубации рекомбинантных TCR-экспрессирующих клеток с клеточной линией плоскоклеточной кациномы UPCI: SCC152 (ATCC® CRL-3240™), антиген-специфической клеточной линией-мишенью, которая представляет собой HPV +, при отношении E:T 7,5:1 или 3,25:1 для клеток, экспрессирующих TCR 16, и при отношении E:T 2,5:1 для клеток, экспрессирующих TCR 49, TCR 53 или TCR 37.
[1026] Результаты показали связывание соответствующего комплекса пептид-МНС-тетрамер, специфичного для каждого TCR. Клетки, экспрессирующие TCR 16, продуцировали IFNγ на уровнях выше фона при обоих протестированных соотношениях E:T. Клетки CD8+, экспрессирующие TCR 49, TCR 53 или TCR 37, продуцировали IFNγ на уровнях выше фона, а клетки CD4+, экспрессирующие TCR 53 и TCR 37, продуцировали IFNγ на уровнях выше фона, что согласуется с CD8-независимой функцией этих TCR в первичных Т-клетках. Результаты согласуются с экспрессией, экспрессией на клеточной поверхности и антиген-специфической функцией рекомбинантных TCR в первичных Т-клетках.
Пример 7: Оценка первичных Т-клеток с помощью нокаута эндогенного гена TCR
[1027] Первичные человеческие CD4+ и CD8+ Т-клетки были выделены и сконструированы для введения генетического нарушения, чтобы нокаутировать (КО) локусы эндогенного гена, которые кодируют цепи Т-клеточного рецептора альфа (TCRα) и бета (TCRβ), или оба с помощью CRISPR/Cas9-опосредованного редактирования генов. Первичные CD4+ и CD8+ Т-клетки от двух здоровых людей-доноров были выделены путем иммуноаффинного отбора из мононуклеарных клеток периферической крови человека (РВМС), полученных от здоровых доноров. Выделенные CD4+ и CD8+ клетки стимулировали в течение 72 часов при 37°C путем культивирования с анти-CD3 /анти-CD28 реагентом при соотношении гранулы:клетки 1:1 в среде, содержащей человеческую сыворотку, IL-2, IL-7 и IL-15.
[1028] Для введения генетического нарушения в локусы TCR реагент анти-CD3/анти-CD28 удаляли, и клетки электропорировали с помощью либо 2 мкМ комплексов рибонуклеопротеина (RNP), содержащих Cas9 Streptococcus pyogenes и гид-РНК (гРНК), нацеленную на ген константной области TCR α (TRAC) или 2 мкМ комплексов RNP, содержащих Cas9 S. pyogenes и гРНК, нацеленную на ген константной области TCR β (TRBC). Для нацеливания как на TRAC, так и на TRBC смесь комплексов RNP подвергали электропорации. В частности, локус TRAC являлся мишенью для генетического нарушения с помощью гРНК с последовательностью нацеливающего домена GAGAAUCAAAAUCGGUGAAU (SEQ ID NO: 1048; последовательность сайта-мишени для TRAC, представленная в SEQ ID NO: 1182), которая нацелена на генетическое нарушение в экзоне 1 эндогенного гена константной области TCR α (TRAC). Локусы TRBC являлись мишенью для генетического нарушение с помощью консенсусной последовательности сайта-мишени, общей для экзона 1 обеих константных областей 1 и 2 TCR β с последовательностью нацеливающего домена GGCCUCGGCGCUGACGAUCU (SEQ ID NO: 1053; последовательность сайта-мишени для TRBC представлена в SEQ ID NO: 1054). В качестве контроля клетки обрабатывали в тех же условиях, которые использовали для электропорации, но без добавления RNP (иммитация).
[1029] Эффективность нокаута измеряли путем оценки экспрессии CD3 с использованием проточной цитометрии через 5 дней после трансфекции, и определяли процент CD3- популяции. Как показано на ФИГ. 7А и 7В, клетки, подвергнутые электропорации с помощью RNP, нацеленных на эндогенные гены TCR, чтобы нарушить эндогенные локусы генов, которые кодируют константную область Т-клеточного рецептора альфа (TCRα) и константную область Т-клеточного рецептора бета (TCRβ), или оба, привели к получению клеток, которые не экспрессируют CD3, что указывает на что они были нокаутированы по экспрессии эндогенного TCR. В этом исследовании приблизительно 80% клеток были нокаутированы по эндогенному TCR, как определено по экспрессии CD3, когда либо TRAC, либо TRBC подвергался направленному воздействию индивидуально, причем большее количество клеток было нокаутировано по эндогенному TCR, когда оба и TRAC, и TRBC подвергались направленному воздействию.
Пример 8. Экспрессия иллюстративных рекомбинантных T-клеточных рецепторов (TCR) в первичных T-клетках с нокаутом эндогенного TCR.
[1030] Первичные человеческие Т-клетки нокаутировали по эндогенным генам, кодирующим Т-клеточный рецептор, с использованием редактирования генов, опосредованного CRISPR/Cas9, и трансдуцировали лентивирусными векторными частицами, кодирующими иллюстративные рекомбинантные E6- или E7-специфичные TCR.
A. Нокаут эндогенных генов TCR и экспрессия рекомбинантного TCR в первичных Т-клетках
[1031] Приблизительно 5×106 первичных человеческих CD4+ и CD8+ T-клеток выделяли путем иммуноаффинного отбора из мононуклеарных клеток периферической крови человека (PBMC), полученных от здоровых доноров. Клетки стимулировали в течение 24 часов путем культивирования анти-CD3/анти-CD28 реагента в среде, содержащей человеческую сыворотку, интерлейкин-2 (IL-2), IL-7 и IL-15 при 37°C до лентивирусной трансдукции. Стимулированные клетки трансдуцировали лентивирусным препаратом, кодирующим TCR 16 (кодон-оптимизированный и цистеин-модифицированный), специфичным для E6 HPV 16 (29-38); TCR 31 (кодон-оптимизированный и цистеин-модифицированный), специфичным для E7 HPV 16 (11-19), описанный в Примере 4 выше; или контролем ложной трансдукции (клетки, обработанные в тех же условиях, что и при лентивирусной трансдукции, но без добавления лентивируса). Лентивирусные конструкции также содержали последовательности, кодирующие укороченный рецептор в качестве суррогатного маркера для трансдукции и экспрессии, отделенные от рекомбинантных TCR-кодирующих последовательностей последовательностью, кодирующей последовательность пропуска рибосомы T2A. После трансдукции клетки культивировали в течение 36-48 часов в среде, содержащей человеческую сыворотку и IL-2, IL-7 и IL-15.
[1032] Чтобы нокаутировать эндогенные гены TCR, клетки подвергали электропорации смесью комплексов рибонуклеопротеинов (RNP), содержащих гид-РНК (гРНК), предназначенную для нацеливания на сайт-мишень в экзоне 1 гена константной области TCR α (последовательность нацеливающего домена гРНК для TRAC, представленная в SEQ ID NO: 1048: последовательность сайта-мишени для TRAC, представленная в SEQ ID NO: 1182), и комплексы RNP, содержащие гРНК, предназначенную для нацеливания на консенсусную последовательность сайта-мишени, общую для экзона 1 обеих константных областей 1 и 2 TCR β (последовательность нацеливающего домена гРНК для TRBC, представленная в SEQ ID NO: 1053; последовательность сайта-мишени для TRBC, представленная в SEQ ID NO: 1054), и белок Cas9 Streptococcus pyogenes, для опосредованного CRISPR/Cas9 редактирования генов локусов TRAC и TRBC (эндо, TCR KO).
[1033] В качестве контроля клетки обрабатывали в тех же условиях, которые использовали для электропорации, но без добавления RNP (эндо. TCR WT). Затем клетки культивировали в культуральной среде, содержащей цитокины. Через три дня после электропорации с RNP трансдуцированные клетки обогащали путем отбора с использованием антитела, направленного против суррогатного маркера, и культивировали в течение 8 дополнительных дней.
[1034] На 13-й день после трансдукции (11-й день после электропорации) клетки оценивали проточной цитометрией на предмет окрашивания антителом против CD3, антителом против CD8 и меченым тетрамером МНС, образующим комплекс с антигеном, распознаваемым рекомбинантным TCR (пептид E6 или E7 HPV16). Продуцирование цитокинов контролировали после инкубации клеток, экспрессирующих рекомбинантный TCR, с клеточной линией плоскоклеточной карциномы UPCI: SCC152 (ATCC® CRL-3240 ™), антиген-специфической клеточной линией-мишенью, которая представляет собой HPV +, при соотношении E:T 7,5:1, 3,25:1 или 0:1 в течение 48 часов. Секрецию интерферон-гамма (IFNγ), IL-2 и фактора некроза опухоли альфа (TNFα) определяли с помощью ИФА.
B. Результаты
[1035] Эффективность нокаута эндогенного TCR оценивали, определяя процент CD3- популяции. Как показано на ФИГ. 8А, более 98% клеток, подвергнутых электропорации с помощью RNP, нацеленных на эндогенные гены TCR (эндо. TCR KO), не экспрессировал CD3, что указывает на то, что они были нокаутированы по экспрессии эндогенного TCR. Для сравнения, более 98% контрольных клеток (эндо. TCR WT) экспрессировали CD3. Результаты показали, что почти вся популяция клеток была нокаутирована по эндогенным генам, кодирующим TCR.
[1036] Экспрессию рекомбинантного TCR сравнивали в клетках с нокаутом эндогенных генов TCR или без него. Как показано на ФИГ. 8B, в клетках, трансдуцированных для экспрессии TCR 16, процент клеток, которые экспрессировали TCR (оценивали по суррогатному маркеру) и связывали тетрамер пептид-MHC, был увеличен в клетках, нокаутированных по эндогенному TCR (эндо. TCR KO), по сравнению с контрольными клетками (эндо. TCR WT), трансдуцированными для экспрессии TCR16. Результаты согласуются с тем, что нокаут эндогенного TCR в первичных T-клетках приводит к повышению экспрессии рекомбинантного TCR.
[1037] После инкубации с антигенспецифической клеточной линией-мишенью клетки, экспрессирующие рекомбинантные TCR и нокаутированные по эндогенным генам TCR (эндо. TCR KO), продуцировал больше IFNγ по сравнению с контрольными клетками (эндо. TCR WT), экспрессирующими тот же TCR и контроль ложной трансдукции (ФИГ. 8C). Аналогичные результаты наблюдали для продуцирования IL-2 и TNFα.
Пример 9: Оценка экспрессии и функции иллюстративных рекомбинантных T-клеточных рецепторов (TCR) в первичных T-клетках с нокаутом эндогенного гена TCR или без него
[1038] Экспрессию и функцию иллюстративных рекомбинантных E7-специфических TCR оценивали в первичных человеческих T-клетках с нокаутом эндогенных генов TCR или без него, или в которых рекомбинантные E7-специфические TCR экспрессировались в виде химерных TCR с мышиной константной областью.
[1039] Первичные человеческие CD4+ и CD8+ T-клетки трансдуцировали лентивирусными препаратами, кодирующими TCR 49, TCR 53 и TCR 37, каждый из которых специфичен для E7 HPV 16 (11-19) (кодон-оптимизированный и цистеин-модифицированный, как описано выше в Примере 4 выше), и суррогатный маркер с использованием методов по существу как описано в Примере 6. В качестве альтернативного подхода первичные человеческие CD4+ и CD8+ T-клетки трансдуцировали лентивирусными препаратами, кодирующими каждый TCR, где Cα и Cβ рекомбинантного TCR были заменены константными областями из TCR мыши (константная область мыши; последовательность Cα мыши, представленная в SEQ ID NO:1012; последовательность Cβ мыши, представленная в SEQ ID NO: 1013). В качестве контроля клетки обрабатывали в тех же условиях, что и для лентивирусной трансдукции, но без добавления лентивируса (ложная трансдукция).
[1040] Клетки, трансдуцированные TCR 49, TCR 53 или TCR 37 (содержащие человеческую константную область), подвергали электропорации с TRAC- и TRBC-нацеленными RNP CRISPR/Cas9 для нокаута эндогенных генов TCR (эндо. TCR KO), по существу, как описано в Примере 8, или обрабатывали в тех же условиях, которые использовали для электропорации, но без добавления RNP (эндо. TCR WT). Другие примеры трансдуцированных клеток также обрабатывали в тех же условиях для электропорации, но без добавления RNP. Клетки обогащали и культивировали, как правило, как описано в Примере 8 выше.
[1041] На 13-й день после трансдукции (11-й день после электропорации) клетки оценивали проточной цитометрией на предмет окрашивания антителом против CD3, антителом против CD8 и HPV16 E7(11-19) пептид-MHC тетрамерным комплексом. Продуцирование интерферона-гамма оценивали после инкубации рекомбинантных TCR-экспрессирующих клеток с антиген-специфическими клетками-мишенями UPCI: SCC152 при соотношении E:T 2,5:1. Цитолитическую активность оценивали путем инкубации CD4+ или CD8+ клеток, экспрессирующих каждый из рекомбинантных TCR, с антиген-специфическими клетками-мишенями UPCI: SCC152 (стабильно экспрессирующими NucLight Red), и оценивая интегральную интенсивность флуоресценции во времени с использованием автоматизированного микроскопа.
Результаты
[1042] Как показано на ФИГ. 9А (TCR 49), ФИГ. 9D (TCR 53) и ФИГ. 9G (TCR 37), экспрессия TCR, оцененная по связыванию тетрамера E7 (11-19), была выше в клетках с нокаутом эндогенных генов TCR (эндо. TCR KO) по сравнению с клетками, которые сохранили эндогенные гены TCR (эндо. TCR WT). Повышенная экспрессия рекомбинантных TCR в первичных T-клетках человека также была достигнута путем введения TCR в виде химерного TCR, содержащего константную область мыши. В некоторых случаях наблюдаемое CD8-независимое связывание тетрамера также увеличивалось в клетках, в которых TCR экспрессировался с нокаутом эндогенного TCR или экспрессией в виде химерного TCR, содержащего константную область мыши.
[1043] Цитолитическая активность, которую контролировали по уменьшенному световому сигналу NucRed клеток, экспрессирующих TCR, наблюдалась после инкубации с антиген-специфическими клетками-мишенями, как показано на ФИГ. 9B (TCR 49), 9E (TCR 53), 9H (TCR 37) и 9J (TCR 37). Для каждого TCR степень цитолитической активности в целом соответствовала наблюдаемому уровню экспрессии TCR среди различных оцененных TCR-экспрессирующих клеток. В некоторых случаях наблюдалась CD4+ цитолитическая активность, согласующаяся с CD8-независимой активностью TCR.
[1044] Как показано на ФИГ. 9C (TCR 49), 9F (TCR 53), 9I (TCR 37) и 9J (TCR 37), продуцирование интерферона-гамма TCR-экспрессирующими клетками после инкубации с антиген-специфическими клетками-мишенями также была повышена в клетках с нокаутом эндогенных генов TCR или в клетках, в которых TCR экспрессировался в виде химерного TCR с константными областями мыши. В некоторых случаях повышенная экспрессия интерферона-гамма наблюдалась в TCR-экспрессирующих CD4+ клетках, что согласуется с CD8-независимой активностью TCR.
Пример 10. Оценка экспрессии, чувствительности к пептидам и функция иллюстративных рекомбинантных T-клеточных рецепторов (TCR) в первичных T-клетках, с элиминированным эндогенным TCR с помощью CRISPR/Cas9-опосредованного редактирования генов локуса TRAC.
[1045] Первичные человеческие CD4+ и CD8+ T-клетки трансдуцировали лентивирусными препаратами, кодирующими различные иллюстративные TCR, специфичные для E7 HPV 16 (11-19) (кодон-оптимизированные и цистеин-модифицированные, как описано в Примере 4). В качестве контроля клетки обрабатывали в тех же условиях, что и для лентивирусной трансдукции, но без добавления лентивируса (ложная трансдукция). Клетки, трансдуцированные TCR, подвергали электропорации с TRAC-нацеленными CRNPR/Cas9 RNP (содержащими последовательность нацеливающих доменов гРНК для TRAC, представленную в SEQ ID NO: 1048) для нокаутирования эндогенных генов TCR (эндо. TCR KO), или обрабатывали в тех же условиях, которые использовали для электропорации, но без добавления RNP (эндо. TCR +). Другие примеры трансдуцированных клеток также обрабатывали в тех же условиях для электропорации, но без добавления RNP. Клетки обогащали и культивировали по существу, как описано в Примере 8.
[1046] Через 6 дней после трансдукции (через 4 дня после электропорации) клетки оценивали проточной цитометрией на предмет окрашивания меченым тетрамером МНС в комплексе с пептидом Е7 HPV16, распознаваемым TCR. Как показано на ФИГ. 10А, улучшенная экспрессия иллюстративного TCR36 наблюдалась в CD8+ T-клетках с нокаутом эндогенных генов TCR (эндо. TCR KO/LV) по сравнению с клетками, которые сохранили эндогенные гены TCR (эндо. ТКС +/LV). Другие иллюстративные протестированные TCR также экспрессировались на более высоких уровнях, как оценивали по связыванию тетрамера E7 (11-19), в эндогенных TCR KO CD8+ или CD4+ первичных T-клетках по сравнению с первичными T-клетками, которые сохранили эндогенные гены TCR (эндо. TCR +) (ФИГ. 10B).
[1047] Чувствительность к пептиду оценивали в эндогенный TCR+или эндогенный TCR KO CD8+ первичных Т-клетках, которые были трансдуцированы для экспрессии иллюстративных TCR, путем титрования пептида E7 (11-19) на клетках T2 и оценки продуцирования цитокинов TCR-экспрессирующими клетками. Приблизительно через 24 часа после инкубации рекомбинантных TCR-экспрессирующих клеток с клетками-мишенями T2, стимулированными с различными концентрациями антигенного пептида, в супернатанте измеряли гамма-интерферон. Как показано на ФИГ. 11А, нокаут эндогенного TCR в первичных человеческих CD8+_T-клетках, сконструированных с помощью иллюстративного TCR 36, улучшал чувствительность к пептиду рекомбинантного TCR, что согласуется с повышенной экспрессией TCR в клетках. Другие иллюстративные TCR также демонстрировали улучшенную чувствительность к пептиду, основанную на продуцировании интерферона-гамма после инкубации с клетками, стимулированными пептидом T2 с концентрацией пептида 10 нг/мл, при экспрессии в эндогенный TCR KO CD8+ первичных T-клетках человека по сравнению с эндогенный TCR+ CD8+ первичными T-клетками человека (ФИГ. 11В).
[1048] Для оценки функции рекомбинантных TCR цитолитическую активность оценивали путем инкубации рекомбинантных TCR-экспрессирующих эффекторных клеток с клетками-мишенями, экспрессирующими E7 HPV 16 при соотношении эффектора к мишени (E:T), равном 5:1. Лизис клеток-мишеней измеряли по потере мишени каждые 2 часа с использованием визуализации живых клеток. Как показано на ФИГ. 12A, в CD8+ T-клетках, экспрессирующих иллюстративный TCR 36, повышенный лизис клеток-мишеней наблюдался в эндогенный TCR KO CD8+ первичных T-клетках по сравнению с эндогенный TCR+ CD8+ первичными T-клетками. Другие иллюстративные TCR также проявляли большую цитолитическую активность при экспрессии в эндогенный TCR KO CD8+ первичных T-клетках по сравнению с CD8+ первичными T-клетками, которые сохранили эндогенные гены TCR (эндо. TCR +) (ФИГ. 12B). Продуцирование цитокина интерферона-гамма, отслеживаемое после инкубации рекомбинантных TCR-экспрессирующих CD8+ эффекторных клеток с клетками-мишенями, экспрессирующими E7 HPV 16 при соотношении E:T 5:1 в течение 48 часов, также было выше в рекомбинантных TCR-экспрессирующих клетках с нокаутом эндогенных генов TCR (эндо. TCR KO) по сравнению с клетками, которые сохранили эндогенные гены TCR (эндо. TCR +) (ФИГ. 12C).
[1049] Результаты согласуются с наблюдением, что во многих иллюстративных протестированных рекомбинантных TCR нокаут эндогенного TCR приводит к повышенной экспрессии, чувствительности к пептиду и функции иллюстративных рекомбинантных TCR.
Пример 11. Противоопухолевый ответ, опосредованный CD4+ клетками, экспрессирующими рекомбинантный TCR.
[1050] Противоопухолевый ответ CD4+ и CD8+ клеток, экспрессирующих рекомбинантный TCR, оценивали на мышиной модели.
A. Противоопухолевый ответ
[1051] Мышиную модель с опухолевыми клетками получали путем подкожной инъекции клеток линии плоскоклеточной карциномы UPCI: SCC152 (ATCC® CRL-3240 ™). Первичные CD4+ клетки, CD8+ клетки или смесь CD4+ и CD8+ клеток, трансдуцированных вектором, кодирующим эталонный TCR, способный связываться с HPV Е7 (11-19) (описано в международной публикации РСТ № WO 2015/184228; эталонный TCR содержит константную область мыши), вводили через 39 дней после введения опухолевых клеток. Средний объем опухоли оценивали с течением времени, вплоть до 63 дней после инъекции опухоли.
[1052] Как показано на ФИГ. 13, объем опухоли уменьшался у мышей, которым вводили только CD4+клетки, экспрессирующие рекомбинантный эталонный TCR (перевернутый треугольник), CD8+ клетки, экспрессирующие рекомбинантный эталонный TCR (треугольник) или смесь CD4+ и CD8+ клеток, экспрессирующих рекомбинантный эталонный TCR (квадрат) по сравнению с мышами, которые не получали никакой обработки (круг). Эти результаты согласуются с наблюдением, что введение только CD4+ клеток, экспрессирующих рекомбинантный TCR, вносит вклад в противоопухолевый ответ, хотя эффект от введения комбинации CD4+ и CD8+ клеток, экспрессирующих рекомбинантный TCR, был выше.
Пример 12: Генерация и оценка сконструированных Т-клеток, экспрессирующих рекомбинантный Т-клеточный рецептор (TCR), путем направленного нокина или случайной интеграции последовательностей, кодирующих TCR
[1053] Полинуклеотиды, кодирующие иллюстративные рекомбинантные Т-клеточные рецепторы (TCR), были введены в Т-клетки с генетическим нарушением в эндогенных локусах генов, которые кодируют альфа-цепь Т-клеточного рецептора (TCRα), с помощью опосредованного CRISPR/Cas9 редактирования генов и направленной интеграции в сайт генетического нарушение посредством зависимой от гомологии репарации (HDR) или путем случайной интеграции посредством лентивирусной трансдукции.
А. Трансгенные конструкции, содержащие рекомбинантный TCR
[1054] Иллюстративные матричные полинуклеотиды были получены для направленной интеграции посредством HDR содержащих трансген последовательностей нуклеиновых кислот, кодирующих один из двух иллюстративных рекомбинантных TCR. Общая структура иллюстративных матричных полинуклеотидов была следующей: [5′-плечо гомологии] - [трансгенные последовательности] - [3′-плечо гомологии]. Плечи гомологии включали приблизительно 600 п.н. последовательностей нуклеиновых кислот, гомологичных последовательностям, окружающим сайт-мишень интеграции в экзоне 1 гена константной области человеческого TCR α (TRAC) (5'-последовательность плеча гомологии, представленная в SEQ ID NO: 1343; 3'-последовательность плеча гомологии, представленная в SEQ ID NO: 1344).
[1055] Трансген включал последовательности нуклеиновых кислот, кодирующие α- и β-цепи иллюстративного рекомбинантного TCR, который распознает эпитоп онкопротеина E7 вируса папилломы человека (HPV) 16 (TCR 49), в котором последовательности, кодирующие цепи TCRα и TCRβ, были разделены элементом пропуска рибосомы 2A. Нуклеотидные последовательности, кодирующие TCR 49, также были модифицированы путем оптимизации кодонов и мутации(мутаций), чтобы способствовать образованию ненативной дисульфидной связи в интерфейсе между константными доменами TCR для увеличения спаривания и стабильности TCR, как описано в Примере 4. Ненативная дисульфидная связь стимулировалась путем модификации цепей TCR в остатке 48 в константной области альфа-цепи TCR (Cα) с Thr на Cys и в остатке 57 константной области бета-цепи TCR (Cβ) с Ser на Cys (см. Kuball et al. (2007) Blood, 109: 2331-2338).
[1056] Трансген также включал а) промотор (EF1α) фактора элонгации человека 1 альфа для управления экспрессией рекомбинантных TCR-кодирующих последовательностей (последовательность, представленная в SEQ ID NO: 1345); или b) последовательности, кодирующие элемент пропуска рибосомы P2A (последовательность, представленная в SEQ ID NO: 1346) перед последовательностями, кодирующими рекомбинантный TCR, для управления экспрессией рекомбинантного TCR из эндогенного локуса TCRα при HDR-опосредованной направленной интеграции в рамке в ген константной области TCR α человека (TRAC).
[1057] Для направленной интеграции с помощью HDR были получены векторные конструкции аденоассоциированного вируса (AAV), содержащие описанные выше матричные полинуклеотиды. Материал AAV получали путем тройной трансфекции вектора AAV, который включал матричный полинуклеотид, плазмиды-помощника серотипа и аденовирусной плазмиды-помощника в клеточную линию 293T. Трансфецированные клетки собирали, лизировали и собирали материал AAV для трансдукции клеток.
[1058] В качестве контроля, для случайной интеграции, последовательности нуклеиновой кислоты, кодирующие иллюстративные трансгенные конструкции рекомбинантного TCR, описанные выше, или последовательности, кодирующие эталонный TCR, способный связываться с E7 HPV 16, но содержащий области Cα и Cβ мыши (описанные в международной публикации PCT No. WO 2015/184228), под контролем промотора EF1α, были включены в иллюстративный лентивирусный вектор, полученный из HIV-1. Псевдотипированные частицы лентивирусного вектора получали стандартными процедурами путем транзиторной трансфекции клеток HEK-293T полученными векторами, плазмидами-помощниками (содержащими плазмиды gagpol и плазмиду rev) и плазмидой псевдотипирования и использовали для трансдукции клеток.
B. Генерация сконструированных Т-клеток
[1059] Первичные CD4+ и CD8+ Т-клетки человека от донора-человека выделяли путем иммуноаффинного отбора из мононуклеарных клеток периферической крови человека (РВМС), полученной от здоровых доноров. Выделенные CD4+ и CD8+ клетки стимулировали в течение 72 часов при 37°C путем культивирования с анти-CD3 /анти-CD28 реагентом при соотношении гранулы:клетки 1:1 в среде, содержащей человеческую сыворотку, IL-2, IL-7 и IL-15. Для введения генетического нарушения в эндогенный локус TRAC с помощью CRISPR/Cas9-опосредованного редактирования генов реагент анти-CD3/анти-CD28 удаляли и клетки подвергали электропорации с помощью 2 мкМ комплексов рибонуклеопротеина (RNP), содержащих Cas9 Streptococcus pyogenes и гид-РНК (гРНК) с последовательностью нацеливающего домена GAGAAUCAAAAUCGGUGAAU (SEQ ID NO: 1048), которая нацелена на генетическое нарушение в экзоне 1 эндогенного гена константной области TCR α (TRAC). После электропорации клетки смешивали со средой, содержащей препарат AAV, содержащий матричный полинуклеотид HDR, кодирующий иллюстративный рекомбинантный TCR, под контролем промотора EF1α для трансдукции (HDR KO). В качестве контроля клетки обрабатывали в тех же условиях, которые использовались для электропорации, но без добавления RNP (ложный КО), трансдуцировали лентивирусным вектором, кодирующим рекомбинантный TCR (Lenti), или эталонным TCR, способным связываться с E7 HPV 16, но содержащим мышиные области Cα и Cβ (Lenti Ref), или трансдуцировали лентивирусным вектором, кодирующим рекомбинантный TCR, а также подвергали электропорации комплексами RNP, нацеленными на генетическое нарушение в локусе TRAC (Lenti KO). После трансдукции клетки культивировали в течение приблизительно 7 дней в средах, содержащих человеческую сыворотку и IL-2, IL-7 и IL-15.
B. Экспрессия TCR
[1060] На 7-й день после электропорации клетки оценивали проточной цитометрией на предмет окрашивания антителом против CD3, антителом против CD4, антителом против CD8, антителом против Vbeta22, специфичным для рекомбинантного TCR, и тетрамером пептид-MHC в комплексе с пептидом E7 HPV 16.
[1061] Результаты для CD8+ клеток, экспрессирующих TCR 49, показаны на ФИГ. 17A-17C. Как показано, клетки, экспрессирующие TCR 49 посредством HDR-опосредованной направленной интеграции в локусе TRAC, демонстрируют самую высокую долю клеток, связанных тетрамером (ФИГ. 17A), и самую высокую среднюю интенсивность флуоресценции окрашивания тетрамера в CD8+ клетках (ФИГ. 17B). Среди CD8+ клеток, связанных с тетрамером, степень связывания с тетрамером, как правило, была более равномерной в клетках, подвергнутых HDR-опосредованной интеграции, по сравнению со случайной интеграцией с помощью лентивирусной трансдукции, о чем свидетельствует более низкий коэффициент вариации (стандартное отклонение сигнала в популяции клеток, деленное на среднее значение сигнала в соответствующей популяции; см. ФИГ. 17C).
[1062] В другом эксперименте с использованием по существу того же метода, который описан выше, окрашивание на TCR с использованием антитела против Vbeta22, специфичного для рекомбинантного TCR или тетрамера пептид-MHC, продемонстрировало более высокую экспрессию TCR49 в первичных человеческих CD8+ T-клетках, которые были нокаутированы по эндогенному TCR либо путем трансдукции лентивирусным вектором, кодирующим рекомбинантный TCR, и электропорацией с комплексами RNP, нацеленными на генетическое нарушение в локусе TRAC (Lenti KO), или путем сайт-направленной вставки рекомбинантного TCR в локус TRAC (HDR KO) (ФИГ.18А). Оценка плотности, измеренная с использованием метода Bangs Lab, включала мечение одноцветных эталонных стандартов флуоресценции для проточной цитометрии теми же флуорофорами, которые использовались для мечения клеток, и создание калибровочной кривой для привязки значений канала флуоресценции к стандартизированным единицам интенсивности флуоресценции. Плотность рецепторов определяли относительно калибровочной кривой. Оценка плотности на основе тетрамера (ФИГ. 18B) или Vβ22 + (ФИГ. 18C) продемонстрировала увеличение плотности TCR на Т-клетках, в которых рекомбинантный TCR был введен путем сайт-направленной вставки рекомбинантного TCR с помощью HDR (HDR KO).
C. Цитолитическая активность и продуцирование цитокинов
[1063] Цитолитическую активность и продуцирование цитокинов клетками, сконструированными для экспрессии рекомбинантного TCR 49 посредством HDR или путем случайной интеграции, как описано выше, оценивали после инкубации с клетками-мишенями, экспрессирующими E7 HPV 16 in vitro.
[1064] Цитолитическую активность оценивали путем культивирования рекомбинантных TCR-экспрессирующих эффекторных клеток с клетками-мишенями, экспрессирующими E7 HPV 16, меченными NucLight Red (NLR), при соотношении эффектор/мишень (E:T) 10:1, 5:1 и 2,5:1. Способность Т-клеток к антиген-специфическому лизису клеток-мишеней оценивали путем измерения потери меченых клеток-мишеней каждые 2 часа до 44 часов после совместного культивирования. Цитолитическую активность определяли по среднему значению от 2 доноров в каждой группе, площади под кривой (AUC) % уничтожения, нормализованной к экспрессии Vbeta, и сравнивали с контролем ложной трансдукции. Продуцирование цитокинов измеряли после инкубации эффекторных клеток, экспрессирующих рекомбинантный TCR, с клетками-мишенями. Секрецию интерферона-гамма (IFNγ) и интерлейкина-2 (IL-2) в супернатанте определяли с помощью ИФА, нормализовали к экспрессии Vbeta и усредняли для 2 доноров в каждой группе.
[1065] Результаты для TCR 49 показаны на ФИГ. 19-20 на основе результатов от двух независимых доноров. Степень уничтожения и секреция IFNγ была выше в клетках, в которые рекомбинантный TCR 49, управляемый промотором EF1α, был введен посредством HDR-опосредованной направленной интеграции в локус TRAC (EF1α-TCR 49 HDR KO), по сравнению со случайной интеграцией, с (TCR 49 Lenti) или без нокаута локуса TRAC (TCR 49 Lenti KO).
E. Вывод
[1066] В целом результаты согласуются с наблюдением, что в некоторых случаях направленная интеграция с помощью HDR последовательностей нуклеиновых кислот, кодирующих иллюстративный рекомбинантный TCR, приводила к более высокой экспрессии рекомбинантного человеческого TCR в Т-клетках человека по сравнению с введением TCR путем случайной интеграции, тем самым приводя к более высокой функциональной активности клеток, экспрессирующих рекомбинантный TCR.
Пример 13: Генерация и оценка экспрессии рекомбинантных T-клеточных рецепторов (TCR) с помощью направленного нокина или случайной интеграции последовательностей, кодирующих TCR, в T-клетках с нокаутом эндогенных цепей TCRα и TCRβ
[1067] Полинуклеотид, кодирующий один из примеров рекомбинантного TCR, был подвергнут направленному воздействию для интеграции в один из сайтов генетического нарушения посредством зависимой от гомологии репарации (HDR) или был введен путем случайной интеграции посредством лентивирусной трансдукции в клетки, сконструированные для генетического нарушения эндогенных локусов генов, которые кодируют цепи альфа (TCRα) или бета (TCRβ) Т-клеточного рецептора или их обе, путем редактирования генов, опосредованного CRISPR/Cas9.
[1068] Для направленной интеграции с помощью HDR препараты AAV, содержащие матричные полинуклеотидные конструкции, кодирующие иллюстративный рекомбинантный TCR 49, были получены по существу, как описано в Примере 10, за исключением следующих отличий: была создана дополнительная конструкция AAV, содержащая промотор MND (последовательность, представленная в SEQ ID NO: 1347), который является синтетическим промотором, который содержит область U3 модифицированного LTR MoMuLV с энхансером вируса миелопролиферативной саркомы для контроля экспрессии последовательностей, кодирующих рекомбинантный TCR.
[1069] Для случайной интеграции лентивирусные препараты, содержащие последовательности нуклеиновых кислот, кодирующие иллюстративный рекомбинантный TCR 49 (кодон-оптимизированный и цистеин-модифицированный, как описано в Примере 4; Lenti человека), были получены в целом, как описано в Примере -10. Для этих исследований конструкция лентивирусной трансдукции дополнительно содержала полинуклеотид, кодирующий укороченный рецептор, отделенный от рекомбинантного трансгена TCR последовательностью, кодирующей последовательность пропуска рибосомы T2A, для экспрессии как рекомбинантного TCR, так и укороченного рецептора из одной и той же конструкции; укороченный рецептор использовался в качестве суррогатного маркера для трансдукции. В качестве контроля был получен полинуклеотид, кодирующий химерный TCR, где Cα и Cβ рекомбинантного TCR были заменены константными областями из TCR мыши (последовательность Cα мыши, представленная в SEQ ID NO: 1012; последовательность Cβ мыши, представленная в SEQ ID NO: 1013; Lenti мыши) или клетки подвергались ложной трансдукции (ложная трансд.).
[1070] Первичные человеческие CD4+ и CD8+ T-клетки были выделены и сконструированы для введения генетического нарушения в эндогенных локусах TRAC и TRBC с помощью CRISPR/Cas9-опосредованного редактирования генов и трансдуцированы препаратами AAV, содержащими матричные полинуклеотиды для HDR, или лентивирусными препаратами для случайной интеграции, в целом как описано в Примере 10B выше, за исключением того, что клетки также были также разрушены в эндогенном локусе TRBC (TRAC/TRBC KO, взаимозаменяемо обозначаемый как TCRαβ KO). Клетки подвергали электропорации с помощью рибонуклеопротеиновых (RNP) комплексов, содержащих гид-РНК, нацеленную на TRAC (гРНК), описанную в примере 10B (содержащую последовательность нацеливающего домена, представленную в SEQ ID NO: 1048), и RNP, содержащих гРНК, нацеленную на консенсусную последовательность сайта-мишени, общую для экзона 1 обеих константных областей 1 и 2 TCR β (с последовательностью нацеливающего домена GGCCUCGGCGCUGACGAUCU (SEQ ID NO: 1053)). После электропорации клетки смешивали со средой, содержащей препараты AAV для трансдукции иллюстративного TCR 49 с помощью HDR, под контролем промотора EF1α (TCRαβ KO/HDR EF1α) или промотора MND (TCRαβ KO/HDR MND).
[1071] В качестве контроля выделенные первичные CD4+ и CD8+ Т-клетки трансдуцировали лентивирусным препаратом, кодирующим иллюстративный рекомбинантный TCR 49 (TCRαβ WT/Lenti человека) или химерный рецептор, содержащий константную область мыши (TCRαβ WT/Lenti мыши), и обрабатывали при тех же условиях, используемых для электропорации, но без добавления RNP (Mock KO; также обозначается как TCRαβ WT или eletro ctrl), или трансдуцировали лентивирусным препаратом, кодирующим иллюстративный рекомбинантный TCR 49, и подвергали электропорации с комплексами RNP, чтобы нокаутировать локусы TRAC и TRBC (TCRαβ KO/Lenti человека).
[1072] Затем клетки культивировали в течение четырех (4) дней, затем оценивали проточной цитометрией на предмет окрашивания антителом против CD3, антителом против Vbeta22, которое распознает рекомбинантный TCR 49, и тетрамером пептид-МНС, образующим комплекс с антигеном, распознаваемым рекомбинантный TCR (пептид E7 HPV 16). Клетки также окрашивали на CD8 или CD4.
[1073] Результаты показаны на ФИГ. 21A-21F. Как показано на ФИГ. 21A и 21B, опосредованный CRISPR/Cas9 нокаут (KO) TRAC и TRBC (панель, обозначенная «TCRαβ KO» на фигурах) приводил к почти полному нарушению экспрессии TCR в CD8+ клетках, что наблюдалось по отсутствию окрашивания CD3 в клетках, подвергнутых КО и ложной трансдукции (панель, обозначенная TCRαβ KO/mock transd.). Экспрессия рекомбинантного TCR (что выявлено по клетками, окрашенными антителом против Vbeta22, или по клеткам, положительным по окрашиванию тетрамерами среди CD8+ клеток), была немного улучшена после лентивирусной трансдукции в клетках, которые были KO по эндогенному TCR (TCRαβ KO/lenti человека) по сравнению с клетками, которые сохраняется экспрессия эндогенного TCR (TCRαβ WT/lenti человека). В клетках, сохраняющих эндогенный TCR, экспрессия рекомбинантного TCR была улучшена лентивирусной трансдукцией рекомбинантного TCR, содержащего константный домен мыши, по сравнению с лентивирусной трансдукцией полностью человеческого рекомбинантного TCR (сравните TCRαβ WT/lenti человека и TCRαβ WT/lenti мыши).
[1074] Опосредованный HDR направленный нокин рекомбинантного TCR и КО эндогенного TCR приводило к значительному увеличению доли клеток, экспрессирующих рекомбинантный TCR, по сравнению с наблюдаемой после лентивирусной трансдукции (первые две левые панели обозначены «HDR» по сравнению с TCRαβ KO/lenti человека на ФИГ.21А и 21В). Среднее геометрическое значение флуоресценции (gMFI) экспрессии рекомбинантного TCR, оцененное по окрашиванию Vbeta22 или окрашиванию тетрамера в CD8+ клетках (ФИГ. 21C) или окрашиванию Vbeta22 в CD4+ клетках (ФИГ. 21D), также была существенно выше в клетках, подвергнутых HDR, по сравнению с лентивирусной трансдукцией. Степень экспрессии рекомбинантного TCR с помощью HDR была сходной, независимо от того, находился ли рекомбинантный TCR под контролем промотора EF1α или MND.
[1075] Среди клеток, которые были положительными по экспрессии рекомбинантного TCR, степень экспрессии была обычно более равномерной или более плотной в клетках, которые были подвергнуты HDR-опосредованной направленной интеграции по сравнению со случайной интеграцией лентивирусной трансдукции (см. ФИГ. 21A и 21B). Как показано на ФИГ. 21Е и ФИГ. 21F, наблюдался более низкий коэффициент вариации (стандартное отклонение сигнала в популяции клеток, деленное на среднее значение сигнала в соответствующей популяции) экспрессии рекомбинантного TCR, определяемой связыванием пептид-MHC тетрамера и экспрессией Vbeta22, соответственно, в CD8+ клетках, которые были подвергнуты HDR-опосредованной интеграции по сравнению со случайной интеграцией. Результаты согласуются с обнаружением того, что направленный нокин рекомбинантного TCR в эндогенный локус TCRα в комбинации с нокаутом эндогенных цепей TCRαβ приводит к более высокому и более равномерному уровню экспрессии в популяции клеток, сконструированных для экспрессии рекомбинантного TCR по сравнению с другими методами.
Пример 14. Оценка HDR-опосредованного нокина последовательностей, кодирующих рекомбинантный Т-клеточный рецептор (TCR), в Т-клетках с нокаутом эндогенных цепей TCRα или TCRβ или обеих.
[1076] Для дальнейшей оценки экспрессии рекомбинантного TCR с помощью HDR последовательности нуклеиновых кислот, кодирующие иллюстративный рекомбинантный TCR в локус TRAC, были подвергнуты направленному воздействию для интеграции в клетки, содержащие двойной нокаут локусов TRAC и TRBC, нокаут только локуса TRAC или нокаут только локуса TRBC.
A. Трансгенные конструкции рекомбинантного TCR и генерация сконструированных клеток
[1077] Эти исследования были проведены с использованием AAV (для HDR) и лентивирусных конструкций (для случайной интеграции), кодирующих иллюстративный TCR 49 (кодон-оптимизированный и цистеин-модифицированный, как описано в Примере 4), по существу, как описано в Примерах 10 и 11, за исключением следующего различия: были получены лентивирусные конструкции, содержащие полинуклеотид, кодирующий рекомбинантный TCR, под функциональным контролем промотора EF1α или MND. Лентивирусную конструкцию, содержащую полинуклеотид, кодирующий рекомбинантный TCR 49 под функциональным контролем промотора EF1α, и укороченный рецептор, отделенный от рекомбинантного трансгена TCR последовательностью, кодирующей последовательность пропуска рибосомы T2A, также генерировали для сравнения.
[1078] Для направленной интеграции с помощью HDR первичные человеческие CD4+ и CD8+ T-клетки стимулировали, культивировали и подвергали электропорации с комплексами рибонуклеопротеина (RNP), содержащими только TRAC-нацеленную гРНК, только TRBC-нацеленную гРНК, или смесь обоих RNP, содержащих TRAC-нацеленную гРНК, и RNP, содержащих TRBC-нацеленную гРНК, как правило, как описано в Примерах 10 и 11, после электропорации клетки трансдуцировали препаратами AAV, содержащими полинуклеотиды, которые кодируют рекомбинантный TCR для нацеливания на эндогенный локус TRAC, как правило, как описано выше.
[1079] Для случайной интеграции первичные человеческие CD4+ и CD8+ T-клетки оттаивали, стимулировали и культивировали по существу, как описано в Примерах 10 и 11, с последующей трансдукцией лентивирусным препаратом, который кодировал рекомбинантный TCR. В этом исследовании препараты лентивируса были трансдуцированы в первичные Т-клетки, которые сохранили эндогенный TCR. В качестве контроля клетки обрабатывали в тех же условиях, что и для лентивирусной трансдукции, но без добавления лентивируса (ложная трансдукция).
B. Экспрессия TCR
[1080] Затем клетки культивировали в течение 4-10 дополнительных дней и оценивали с помощью проточной цитометрии после окрашивания антителом против CD3, антителом против Vbeta22, специфичным для рекомбинантного TCR 49, и тетрамером пептид-МНС, образующим комплекс с распознаваемым антигеном с помощью TCR (пептид E7 HPV 16). Клетки также окрашивали на CD8 или CD4.
[1081] Результаты для окрашивания CD3 показаны на ФИГ. 22A-22C, для окрашивания тетрамера показано на ФИГ. 23A-23C, и для окрашивания Vbeta22 показано на ФИГ. 24A-24D.
[1082] Как показано на ФИГ. 22A и 22C, электропорация со смесями RNP в комплексе с гРНК, нацеленными на TRAC, и RNP в комплексе с гРНК, нацеленными на TRBC, приводила к эффективному нокауту эндогенного TCR, о чем свидетельствует отсутствие поверхностной экспрессии CD3 (см. панель, помеченную KO/ложная трансд. на ФИГ. 22А; см. также группу, помеченную KO mock ФИГ. 22C, который показывает процент CD3+CD8+ клеток среди CD8+ клеток). Степень KO с RNP, нацеленными на оба, TRAC и TRBC, была выше, чем для клеток, электропорированных с RNP в комплексе с гРНК, нацеленной только на TRAC или только на TRBC, что согласуется с тем наблюдением, что двойное нацеливание на оба константных домена цепей TCR α и β повышает эффективность нарушения экспрессии эндогенного TCR. В клетках, трансдуцированных лентивирусными векторами, в которых не было нарушений эндогенного TCR, экспрессия CD3 была одинаковой во всех тестируемых условиях (ФИГ. 22B и 22C). Как показано на ФИГ. 22A и 22C, экспрессия CD3 также была сходной среди клеток, в которых рекомбинантный TCR был введен посредством HDR, что согласуется с поверхностной экспрессией TCR/CD3 в клетках, в которые был введен рекомбинантный TCR.
[1083] Как показано на ФИГ. 23A и 23C, доля CD8+ клеток, которые связывают тетрамер пептид-MHC, выявляя на экспрессию рекомбинантного TCR, была выше в условиях, в которых HDR проводился в клетках, нокаутированных по обоим, TRAC и TRBC, по сравнению только с TRAC (сравните верхние и средние ряды на ФИГ.23А; сравните TRAC & TRBC только с TRAC на ФИГ. 23С). Как показано на ФИГ. 23C, аналогичные результаты наблюдались в дни 7 и 13. Подобная экспрессия рекомбинантного TCR наблюдалась в клетках независимо от того, проводили ли HDR с конструкцией для интеграции под контролем экзогенного промотора EF1α или MND или под контролем эндогенного промотора TCR (P2A-содержащая конструкция). Как показано на ФИГ. 23B и ФИГ. 23C, меньшее количество клеток экспрессировало рекомбинантный TCR, согласно оценке окрашиванием тетрамера, после лентивирус-опосредованной трансдукции, независимо от присутствия укороченного рецептора в лентивирусных конструкциях.
[1084] Как показано на ФИГ. 24A-24C, аналогично приведенным выше результатам, экспрессия рекомбинантного TCR на CD8+ T-клетках наблюдалась при прямом окрашивании рекомбинантного TCR антителом, которое специфически распознает цепь Vbeta рекомбинантного TCR. Окрашивание антителом против Vbeta, которое также способно детектировать рекомбинантный TCR на CD4+ T-клетках (CD8-отрицательная популяция), также показало, что экспрессия рекомбинантного TCR наблюдалась в CD4+ клетках (ФИГ. 24A и ФИГ. 24D).
[1085] Для всех методов оценки экспрессии рекомбинантного TCR, показанных выше (анти-CD3, тетрамер и анти-Vbeta22), приведенные выше результаты также показали, что направленная интеграция рекомбинантного TCR в TRAC через HDR специфична для индуцированного нуклеазой разрыва ДНК в TRAC локусе, так как клетки, электропорированные с использованием TRBC-нацеленных RNP, не экспрессируют рекомбинантный TCR (см. условие «только TRBC» на ФИГ.).
C. Цитолитическая активность и продуцирование цитокинов
[1086] Цитолитическую активность и продукцию цитокинов CD8+ клетками, сконструированными для экспрессии рекомбинантного TCR 49 посредством HDR или путем случайной интеграции, как описано выше, оценивали после инкубации с клетками-мишенями, экспрессирующими E7 HPV 16 in vitro. В дополнение к клеткам, описанным выше, также оценивали первичные человеческие CD8+ клетки, трансдуцированные лентивирусом, кодирующим эталонный TCR, способный связываться с E7 HPV 16, но содержащий мышиные области Cα и Cβ.
[1087] Цитолитическую активность оценивали путем инкубации рекомбинантных TCR-экспрессирующих эффекторных клеток с клетками-мишенями, экспрессирующими E7 HPV 16 при соотношении эффектора к мишени (E:T) 10:1, 5:1 и 2,5:1. Способность Т-клеток антигенспецифически лизировать клетки-мишени оценивали через 4 часа после совместного культивирования. Цитолитическую активность определяли по площади под кривой (AUC)% уничтожения, нормализованной к экспрессии Vbeta, и сравнивали с контролем ложной трансдукции. Результаты показаны на ФИГ. 25. Степень уничтожения была выше в клетках, в которые рекомбинантный TCR был введен посредством HDR-опосредованной направленной интеграции, по сравнению со случайной интеграцией, что согласуется с выводом о том, что более высокая экспрессия рекомбинантного TCR в клетках приводит к более высокой функциональной активности.
[1088] Продуцирование цитокинов также контролировали после инкубации CD8+ эффекторных клеток, экспрессирующих рекомбинантный TCR, с клетками-мишенями, экспрессирующими E7 HPV 16, при соотношении E:T 10:1 и 2,5:1 в течение 48 часов. Секрецию IFNγ в супернатанте определяли с помощью ИФА и нормализовали к экспрессии Vbeta22 для каждой группы. Результаты показаны на ФИГ. 26. По аналогии с результатами, приведенными выше для цитолитической активности, наблюдали более высокое продуцирование IFNγ клетками, подвергнутыми HDR-опосредованной интеграции, по сравнению со случайной интеграцией. В анализе цитолитической активности и оценке секреции IFNγ функциональная активность клеток, экспрессирующих рекомбинантный TCR посредством HDR-опосредованной интеграции, была аналогична активности клеток, экспрессирующих эталонный TCR, содержащий константные домены мыши, посредством лентивирусной трансдукции.
[1089] Пролиферацию рекомбинантных TCR-экспрессирующих клеток оценивали после инкубации с клетками-мишенями SCC152 или клетками-мишенями T2, стимулированными антигенным пептидом. Клетки метили фиолетовым красителем CellTrace™ (ThermoFisher). Деление живых Т-клеток было показано разведением фиолетового красителя CellTrace™, что было оценено с помощью проточной цитометрии.
[1090] Результаты различных функциональных анализов представлены на ФИГ. 27. Как показано на тепловой карте, изображающей относительную активность рекомбинантных TCR-экспрессирующих клеточных популяций в различных функциональных активностях (AUC% уничтожения при соотношениях E:T 10:1, 5:1 и 2,5:1 ((обозначено «AUC»), связывании тетрамера в CD8+ клетках в дни 7 и 13 (обозначенное «тетрамер CD8»), анализе пролиферации (обозначено «количество CTV») с использованием клеток SCC152 или клеток-мишеней Т2, стимулированных антигенным пептидом, и секреции IFNγ из CD8+ клеток (обозначено «CD8 секретируемый IFNg»)), функциональная активность клеток с рекомбинантным TCR, являющихся мишенью для нокина в локус константного домена эндогенной цепи TCRα и нокаута эндогенных генов TCRαβ или гена TCRα, как правило, была выше по сравнению с клетками, где полинуклеотид, кодирующий рекомбинантный TCR был случайным образом интегрирован.
[1091] В целом результаты согласуются с наблюдением, что направленная интеграция с помощью HDR приводит к более высокой экспрессии TCR человеческого TCR в человеческих T-клетках по сравнению с введением TCR путем случайной интеграции, тем самым приводя к более высокой функциональной активности клеток, экспрессирующих рекомбинантный TCR.
[1092] Настоящее изобретение не предназначено для ограничения объема конкретными раскрытыми вариантами осуществления, которые предложены, например, для иллюстрации различных аспектов изобретения. Различные модификации описанных композиций и способов станут очевидными из описания и предложенных в настоящем описании идей. Такие вариации могут быть осуществлены без отклонения от истинного объема и сущности раскрытия и подразумевается, что они попадают в объем настоящего раскрытия.
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
P2A
Искусственная
(nt)
P2A
Искусственная
(нт)
P2A
Искусственная
(нт)
Кодон-оптимизированная/Цистеин-модифицированная
Homo sapiens
(нт)
Кодон-оптимизированная/Цистеин-модифицированная
Homo sapiens
(нт)
Кодон-оптимизированная/Цистеин-модифицированная
Homo sapiens
(нт)
Кодон-оптимизированная/Цистеин-модифицированная
Homo sapiens
(нт)
Кодон-оптимизированная/Цистеин-модифицированная
Homo sapiens
(нт)
Кодон-оптимизированная/Цистеин-модифицированная
Homo sapiens
(нт)
Homo sapiens
(нт)
Homo sapiens
(нт)
Кодон-оптимизированная/Цистеин-модифицированная Полноразмерная последовательность
Homo sapiens
(нт)
Нативный
Homo sapiens
(нт)
Нативный
Homo sapiens
(нт)
Нативный
Homo sapiens
(ак)
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(нт)
Кодон-оптимизированная/Цистеин-модифицированная
Homo sapiens
(нт)
Нативный
Homo sapiens
(ак)
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(нт)
Кодон-оптимизированная/Цистеин-модифицированная
Homo sapiens
(нт)
Кодон-оптимизированная/Цистеин-модифицированная Полноразмерная последовательность
Homo sapiens
(нт)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(ак)
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(нт)
Кодон-оптимизированная/Цистеин-модифицированная
Homo sapiens
(нт)
Нативный
Homo sapiens
(ак)
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(нт)
Кодон-оптимизированная/Цистеин-модифицированная
Homo sapiens
(нт)
Кодон-оптимизированная/Цистеин-модифицированная Полноразмерная последовательность
Homo sapiens
(нт)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(ак)
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(нт)
Кодон-оптимизированная/Цистеин-модифицированная
Homo sapiens
(нт)
Нативный
Homo sapiens
(ак)
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(нт)
Кодон-оптимизированная/Цистеин-модифицированная
Homo sapiens
(нт)
Кодон-оптимизированная/Цистеин-модифицированная Полноразмерная последовательность
Homo sapiens
(нт)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(ак)
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(нт)
Кодон-оптимизированная/Цистеин-модифицированная
Homo sapiens
(нт)
Нативный
Homo sapiens
(ак)
Цистеин-модифицированная
Homo sapiens
(ак)
Кодон-оптимизированная/Цистеин-модифицированная
Homo sapiens
(нт)
Нативный
Homo sapiens
(нт)
Кодон-оптимизированная/Цистеин-модифицированная Полноразмерная последовательность
Homo sapiens
(нт)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(ак)
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(нт)
Кодон-оптимизированная/Цистеин-модифицированная
Homo sapiens
(нт)
Нативный
Homo sapiens
(ак)
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(нт)
Кодон-оптимизированная/Цистеин-модифицированная
Homo sapiens
(нт)
Кодон-оптимизированная/Цистеин-модифицированная Полноразмерная последовательность
Homo sapiens
(нт)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(ак)
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(нт)
Кодон-оптимизированная/Цистеин-модифицированная
Homo sapiens
(нт)
Нативный
Homo sapiens
(ак)
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(нт)
Кодон-оптимизированная/Цистеин-модифицированная
Homo sapiens
(нт)
Кодон-оптимизированная/Цистеин-модифицированная Полноразмерная последовательность
Homo sapiens
(нт)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(ак)
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(нт)
Кодон-оптимизированная/Цистеин-модифицированная
Homo sapiens
(нт)
Нативный
Homo sapiens
(ак)
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(нт)
Кодон-оптимизированная/Цистеин-модифицированная
Homo sapiens
(нт)
Кодон-оптимизированная/Цистеин-модифицированная Полноразмерная последовательность
Homo sapiens
(нт)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(ак)
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(нт)
Кодон-оптимизированная/Цистеин-модифицированная
Homo sapiens
(нт)
Нативный
Homo sapiens
(ак)
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(нт)
Кодон-оптимизированная/Цистеин-модифицированная
Homo sapiens
(нт)
Кодон-оптимизированная/Цистеин-модифицированная Полноразмерная последовательность
Homo sapiens
(нт)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(ак)
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(нт)
Кодон-оптимизированная/Цистеин-модифицированная
Homo sapiens
(нт)
Нативный
Homo sapiens
(ак)
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(нт)
Кодон-оптимизированная/Цистеин-модифицированная
Homo sapiens
(нт)
Кодон-оптимизированная/Цистеин-модифицированная Полноразмерная последовательность
Homo sapiens
(нт)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(нт)
Mus musculus
(ак)
Нативный
Homo sapiens
(ак)
Альфа, вариабельная область
Homo sapiens(ак)
Бета, вариабельная область
Homo sapiens
(ак)
Homo sapiens
(ак)
Homo sapiens(ак)
Homo sapiens
(ак)
Homo sapiens
(ак)
Альфа, вариабельная область
Homo sapiens
(ак)
Бета, вариабельная область
Homo sapiens
(ак)
Homo sapiens
(ак)
Homo sapiens
(ак)
Альфа, вариабельная область
Homo sapiens
(ак)
Бета, вариабельная область
Homo sapiens
(ак)
Альфа, вариабельная область
Homo sapiens
(ак)
Бета, вариабельная область
Homo sapiens
(ак)
Альфа, вариабельная область
Homo sapiens
(ак)
Бета, вариабельная область
Homo sapiens
(ак)
Альфа, вариабельная область
Homo sapiens
(ак)
Бета, вариабельная область
Homo sapiens
(ак)
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(ак)
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(ак)
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(ак)
Цистеин-модифицированная
Homo sapiens
(ак)
Homo sapiens
(ак)
Homo sapiens
(ак)
Homo sapiens
(ак)
Homo sapiens
(ак)
Homo sapiens
(ак)
Homo sapiens
(ак)
TCR 11
Альфа CDR1
Homo sapiens
(ак)
TCR 11
Альфа CDR2
Homo sapiens
(ак)
Homo sapiens
(ак)
Homo sapiens
(ак)
Homo sapiens
(ак)
Homo sapiens
(ак)
TCR 10/TCR 14 - TCR Бета CDR1
Homo sapiens
(ак)
Homo sapiens
(ак)
Homo sapiens
(ак)
Альфа CDR1
Homo sapiens
(ак)
Альфа CDR2
Homo sapiens
(ак)
Альфа CDR3
Homo sapiens
(ак)
Homo sapiens
(ак)
Бета CDR2
Homo sapiens
(ак)
Бета CDR3
Homo sapiens
(ак)
Альфа CDR1
Homo sapiens
(ак)
Homo sapiens
Homo sapiens
(ак)
Homo sapiens
(ак)
Альфа CDR1
Homo sapiens
(ак)
Альфа CDR2
Homo sapiens
Альфа CDR3
Homo sapiens
Бета CDR3
Homo sapiens
(ак)
Альфа CDR1
Homo sapiens
(ак)
Альфа CDR2
Homo sapiens
(ак)
Альфа CDR3
Homo sapiens
(ак)
Бета CDR1
Homo sapiens
(ак)
Бета CDR2
Homo sapiens
(ак)
Бета CDR3
Homo sapiens
(ак)
Альфа CDR1
Homo sapiens
(ак)
Альфа CDR2
Homo sapiens
(ак)
Альфа CDR3
Homo sapiens
(ак)
Бета CDR3
Homo sapiens
(ак)
Альфа CDR3
Homo sapiens
(ак)
Бета CDR1
Homo sapiens
(ак)
Бета CDR2
Homo sapiens
(ак)
Бета CDR3
Homo sapiens
(ак)
Нативный
Homo sapiens
(ак)
Цистеин-модифицированная
Homo sapiens
(ак)
TCR Альфа сигнальный пептид
Homo sapiens
(ак)
Homo sapiens
(ак)
Нативный
Homo sapiens
(нт)
Homo sapiens
(ак)
Homo sapiens
(ак)
TCR 8/TCR10/TCR 14 -
TCR Бета сигнальный пептид
Homo sapiens
(ак)
Homo sapiens
(ак)
Homo sapiens
(ак)
Homo sapiens
(ак)
Homo sapiens
(ак)
Homo sapiens
(ак)
Homo sapiens
(ак)
Homo sapiens
(ак)
Homo sapiens
(ак)
Нативный
Homo sapiens
(ак)
TCR 5 - (E6)29/TCR 12 /TCR 55- TCR Альфа, константная область
Homo sapiens
(ак)
Homo sapiens
(ак)
TCR 10
TCR Альфа, константная область
Homo sapiens
(ак)
Homo sapiens
(ак)
TCR 11
Альфа, константная область
Homo sapiens
(ак)
Homo sapiens
(ак)
Нативный
Homo sapiens
(нт)
Homo sapiens
(ак)
Искусственная(ак)
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(ак)
TCR 6/
TCR 8/
TCR 9/
TCR 10
TCR 11
P2A
Искусственная
(нт)
P2A
Искусственная
(нт)
P2A
Искусственная
(нт)
P2A
Искусственная
(нт)
Искусственная
(ак)
TCR 10
Нативный TCR Альфа, константная область
Homo sapiens
(ак)
Homo sapiens
(ак)
Homo sapiens
(ак)
TCR 10
Нативный TCR Альфа, константная область
Homo sapiens
(ак)
Нативный TCR Бета, константная область
Homo sapiens
(ак)
TCR 11
Нативный TCR Альфа, константная область
Homo sapiens
(ак)
Homo sapiens
(ак)
Нативный
Homo sapiens
(нт)
TCR 5/TCR 12 /TCR 8/
TCR 9/TCR 13- (E6)29 - Нативный TCR Альфа, константная область
Homo sapiens
(ак)
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(ак)
Полноразмерная последовательность
Нативный
Homo sapiens
(ак)
Полноразмерная последовательность
Нативный
Homo sapiens
(ак)
Полноразмерная последовательность
Нативный
Homo sapiens
(ак)
Полноразмерная последовательность
Нативный
Homo sapiens
(ак)
Полноразмерная последовательность
Нативный
Homo sapiens
(ак)
Полноразмерная последовательность
Нативный
Homo sapiens
(ак)
Полноразмерная последовательность
Нативный
Homo sapiens
(ак)
Полноразмерная последовательность
Нативный
Homo sapiens
(ак)
Полноразмерная последовательность
Нативный
Homo sapiens
(ак)
X1=T, D, S или N;
X2=I или S;
X3=S, D, N, Y или A;
X4=G, Q, P или отсутствует;
X5=T, S, F или I;
X6=D, Y, P или Q;
X7=Y, G, N, A, S или Q
X1=D или V;
X3=S или P;
X4=S или F;
X5=T или S;
X6=Y или N
Цистеин-модифицированная
Homo sapiens
(ак)
X1=T, D, N, S или V;
X2=I или S;
X3=S, D, A, P, N или Y
X4=G, Q, P или отсутствует;
X5=T, S, I или F;
X6=D, Y, Q, T, P или S;
X7=Y, G, N, A, S или Q;
X1=G, Q, I, M, Y или V;
X2=L, S, Q, T или Y;
X3=T, G, L или S;
X4=Y, S, N, A или отсутствует;
X5=отсутствует, A или D;
X6=отсутствует, E, Q, T или S;
X7=S, Q, R, L или G;
X8=N, V или E;
X1=I или M;
X2=F или T;
X3=S или F;
X4=N или S;
X5=M или E;
X6=D или N;
X7=M или T;
Homo sapiens
(ак)
X1=G, Q, I, V, Y или M;
X2=L, S, Q, Y, F или T;
X3=T, G, S, L или F;
X4=Y, S, N, A или отсутствует;
X5=отсутствует, A или D;
X6=отсутствует, E, Q, S, M или T;
X7=S, Q, R, G, D, L или N;
X8=N, E, M, T или V
X1=A, I или V;
X2=M, L, S или V;
X3=R, L, Q или N;
X4=E, V, T, P, G или F;
X5=G, I, L, A, отсутствует или P;
X6=R, T, G, отсутствует или S;
X7=G, R или отсутствует;
X8=T, G или отсутствует;
X9=отсутствует или A;
X10=отсутствует или G;
X11=отсутствует или G;
X12=отсутствует или T;
X13=отсутствует или S;
X14=G, Y, отсутствует или N;
X15=F, G, N или T;
X16=K или N, P;
X17=T или L;
X18=I, V, F или T
X1=A или V;
X2=E или V;
X3=S или P
X4=I, S или R;
X5=R, G или D;
X6=G, A или N;
X7=F, отсутствует или Y;
X8=G или T
X9=N, T или Q;
X10=V, K или N;
X11=L или F;
X12=H, I или V
Homo sapiens
(ак)
X1=A, I или V;
X2=M, L, V, E или S;
X3=R, L, N, Q, P или S;
X4=E, V, P, T, F, I, R, G, S или A;
X5=G, I, L, A, P, R, D, отсутствует или H;
X6=R, T, G, S, N, отсутствует или A;
X7=G, R, N или отсутствует;
X8=T, G или отсутствует;
X9=отсутствует или A;
X10=отсутствует или G;
X11=отсутствует или G;
X12=отсутствует или T;
X13=F, Y, S или отсутствует;
X14=G, Y, отсутствует или N;
X15=F, G, T, N, Q или Y;
X16=K, P, V, N или A;
X17=T, L или F;
X18=I, V, T, H, F или N
X1=S или M;
X2=G, E, D или N;
X4=V, N или E;
X5=S, R, N или Y;
Нативный
Homo sapiens
(ак)
X1=S или M;
X2=G, E, D, N или Q;
X3=H или V;
X4=V, N, E, L или T;
X5=S, R, N, Y или M;
X1=F или S;
X2=Q, Y или V;
X3=N, D или G;
X4=E или V;
X5=A, K или G;
X6=Q, M или T;
Цистеин-модифицированная
Homo sapiens
(ак)
X1=F, S или A;
X2=Q, Y, V или N;
X3=N, D, G или Q;
X5=E, V, N или S;
X6=A, K, G или E;
X7=Q, M, T, I или A;
X3=S или T
X4=H, P, L, F или Y;
X5=L, G, W, F, T или S;
X6=A, G или L;
X7=G, E, A, T, Q или отсутствует;
X8=F, G, T, R или S;
X9=T, N, H, R, E или A;
X10=G, T, Q, D, R или Y;
X11=E, P, T или G;
X12=L, A, Q или Y;
X13=F, H, Y или T
X2=S или I;
X4=T или D;
X6=S или T;
X7=S или N;
X9=E или G;
X10=Q или Y;
X11=Y или T
Нативный
Homo sapiens
(ак)
X1=A или S;
X2=S, I или V;
X3=S, T или V;
X4=H, P, L, Y, T, D или F;
X5=L, G, W, F, S, T или R;
X6=A, G, L, S или T;
X7=G, E, A, T, R, Q или отсутствует;
X8=отсутствует или G;
X9=отсутствует или G;
X10=отсутствует, F, G, T, S или R;
X11=T, N, H, A, S, R или E;
X12=G, T, Q, D, Y или R;
X13=E, P, T или G;
X14=L, A, Q или Y;
X15=F, H, Y или T
ETDMNLNFQN LSVMGLRILL LKVAGFNLLM TLRLWSS
Mus musculus
(ак)
Mus musculus
(ак)
(ак)
(ак)
Homo sapiens(ак)
Homo sapiens
(нт)
Homo sapiens
(ак)
Homo sapiens
(ак)
Homo sapiens
(ак)
Искусственная
(ак)
Искусственная(ак)
Homo sapiens
(ак)
Homo sapiens
(ак)
Homo sapiens
(ак)
Homo sapiens
(ак)
Homo sapiens
(ак)
Homo sapiens
(ак)
Homo sapiens
(ак)
Homo sapiens
(ак)
Нативный
Homo sapiens
(ак)
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(ак)
Homo sapiens
(ак)
Нативный
Homo sapiens
(ак)
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(ак)
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(ак)
Homo sapiens
(ак)
Homo sapiens
(ак)
Homo sapiens
(ак)
Homo sapiens
(ак)
Homo sapiens
(ак)
Homo sapiens
(ак)
Homo sapiens
(ак)
CDR3 Альфа
Homo sapiens
(ак)
CDR1 Альфа
Homo sapiens
(ак)
CDR2 Альфа
Homo sapiens
(ак)
CDR3 Альфа
Homo sapiens
(ак)
CDR3 Бета
Homo sapiens
(ак)
CDR1 Альфа
Homo sapiens
(ак)
CDR2 Альфа
Homo sapiens
(ак)
CDR3 Альфа
Homo sapiens
(ак)
CDR3 Бета
Homo sapiens
(ак)
Сигнальная последовательность
Homo sapiens
(ак)
Сигнальная последовательность
Homo sapiens
(ак)
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(ак)
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(ак)
Цистеин-модифицированная
Homo sapiens
(ак)
Mus musculus
(ак)
Нативный
Homo sapiens
(ак)
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(ак)
Цистеин-модифицированная
Homo sapiens
(ак)
Homo sapiens
(ак)
Homo sapiens
(ак)
Нативный
Homo sapiens
(ак)
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(ак)
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(ак)
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(ак)
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(ак)
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(ак)
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(ак)
Цистеин-модифицированная
Homo sapiens
(ак)
Нативный
Homo sapiens
(ак)
Цистеин-модифицированная
Homo sapiens
(ак)
Полноразмерная последовательность
Нативный
Homo sapiens
(ак)
Полноразмерная последовательность
Нативный
Homo sapiens
(ак)
Полноразмерная последовательность
Нативный
Homo sapiens
(ак)
Искусственная
Искусственная (нт)
NCBI Эталонная последовательность: NG_001332.3, TRAC
NCBI Эталонная последовательность: NG_001333.2, TRBC1
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
Полноразмерная последовательность
Цистеин-модифицированная
Homo sapiens
(ак)
--->
СПИСОК ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> Juno Therapeutics, Inc.
Brandt, Cameron
<120> HPV-СПЕЦИФИЧЕСКИЕ СВЯЗЫВАЮЩИЕ МОЛЕКУЛЫ
<130> 735042014140
<140> Not Yet Assigned
<141> Concurrently Herewith
<150> 62/567750
<151> 2017-10-03
<150> 62/597411
<151> 2017-12-11
<150> 62/653529
<151> 2018-04-05
<160> 1391
<170> FastSEQ для Windows версии 4.0
<210> 1
<211> 606
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 14 Полноразмерная последовательность, цистеин-модифицированная
<400> 1
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Thr Phe Trp Gly Gln Arg Arg Thr Glu Ala Phe Phe Gly Gln Gly Thr
115 120 125
Arg Leu Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu
305 310 315 320
Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Glu Lys Asn
325 330 335
Pro Leu Ala Ala Pro Leu Leu Ile Leu Trp Phe His Leu Asp Cys Val
340 345 350
Ser Ser Ile Leu Asn Val Glu Gln Ser Pro Gln Ser Leu His Val Gln
355 360 365
Glu Gly Asp Ser Thr Asn Phe Thr Cys Ser Phe Pro Ser Ser Asn Phe
370 375 380
Tyr Ala Leu His Trp Tyr Arg Trp Glu Thr Ala Lys Ser Pro Glu Ala
385 390 395 400
Leu Phe Val Met Thr Leu Asn Gly Asp Glu Lys Lys Lys Gly Arg Ile
405 410 415
Ser Ala Thr Leu Asn Thr Lys Glu Gly Tyr Ser Tyr Leu Tyr Ile Lys
420 425 430
Gly Ser Gln Pro Glu Asp Ser Ala Thr Tyr Leu Cys Ala Ser Gln Thr
435 440 445
Gly Ala Asn Asn Leu Phe Phe Gly Thr Gly Thr Arg Leu Thr Val Ile
450 455 460
Pro Tyr Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser
465 470 475 480
Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln
485 490 495
Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys
500 505 510
Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val
515 520 525
Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn
530 535 540
Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys
545 550 555 560
Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn
565 570 575
Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val
580 585 590
Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600 605
<210> 2
<211> 604
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 13 Полноразмерная последовательность, цистеин-модифицированная
<400> 2
Met Gly Thr Arg Leu Leu Cys Trp Val Val Leu Gly Phe Leu Gly Thr
1 5 10 15
Asp His Thr Gly Ala Gly Val Ser Gln Ser Pro Arg Tyr Lys Val Ala
20 25 30
Lys Arg Gly Gln Asp Val Ala Leu Arg Cys Asp Pro Ile Ser Gly His
35 40 45
Val Ser Leu Phe Trp Tyr Gln Gln Ala Leu Gly Gln Gly Pro Glu Phe
50 55 60
Leu Thr Tyr Phe Gln Asn Glu Ala Gln Leu Asp Lys Ser Gly Leu Pro
65 70 75 80
Ser Asp Arg Phe Phe Ala Glu Arg Pro Glu Gly Ser Val Ser Thr Leu
85 90 95
Lys Ile Gln Arg Thr Gln Gln Glu Asp Ser Ala Val Tyr Leu Cys Ala
100 105 110
Ser Ser Pro Thr Gly Thr Glu Arg Glu Leu Phe Phe Gly Glu Gly Ser
115 120 125
Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser
305 310 315 320
Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Leu
325 330 335
Leu Leu Leu Val Pro Val Leu Glu Val Ile Phe Thr Leu Gly Gly Thr
340 345 350
Arg Ala Gln Ser Val Thr Gln Leu Asp Ser His Val Ser Val Ser Glu
355 360 365
Gly Thr Pro Val Leu Leu Arg Cys Asn Tyr Ser Ser Ser Tyr Ser Pro
370 375 380
Ser Leu Phe Trp Tyr Val Gln His Pro Asn Lys Gly Leu Gln Leu Leu
385 390 395 400
Leu Lys Tyr Thr Ser Ala Ala Thr Leu Val Lys Gly Ile Asn Gly Phe
405 410 415
Glu Ala Glu Phe Lys Lys Ser Glu Thr Ser Phe His Leu Thr Lys Pro
420 425 430
Ser Ala His Met Ser Asp Ala Ala Glu Tyr Phe Cys Val Val Arg Gly
435 440 445
Gly Lys Leu Ile Phe Gly Gln Gly Thr Glu Leu Ser Val Lys Pro Asn
450 455 460
Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser
465 470 475 480
Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn
485 490 495
Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys Val
500 505 510
Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp
515 520 525
Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile
530 535 540
Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val
545 550 555 560
Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln
565 570 575
Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly
580 585 590
Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600
<210> 3
<211> 606
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 12 / TCR 55 Полноразмерная последовательность, цистеин-модифицированная
<400> 3
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ser Thr Thr Arg Ser Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser Leu
305 310 315 320
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Lys Thr
325 330 335
Phe Ala Gly Phe Ser Phe Leu Phe Leu Trp Leu Gln Leu Asp Cys Met
340 345 350
Ser Arg Gly Glu Asp Val Glu Gln Ser Leu Phe Leu Ser Val Arg Glu
355 360 365
Gly Asp Ser Ser Val Ile Asn Cys Thr Tyr Thr Asp Ser Ser Ser Thr
370 375 380
Tyr Leu Tyr Trp Tyr Lys Gln Glu Pro Gly Ala Gly Leu Gln Leu Leu
385 390 395 400
Thr Tyr Ile Phe Ser Asn Met Asp Met Lys Gln Asp Gln Arg Leu Thr
405 410 415
Val Leu Leu Asn Lys Lys Asp Lys His Leu Ser Leu Arg Ile Ala Asp
420 425 430
Thr Gln Thr Gly Asp Ser Ala Ile Tyr Phe Cys Ala Val Pro Ser Gly
435 440 445
Ala Thr Asn Lys Leu Ile Phe Gly Thr Gly Thr Leu Leu Ala Val Gln
450 455 460
Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser
465 470 475 480
Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln
485 490 495
Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys
500 505 510
Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val
515 520 525
Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn
530 535 540
Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys
545 550 555 560
Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn
565 570 575
Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val
580 585 590
Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600 605
<210> 4
<211> 66
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCR 14 P2A
<400> 4
ggctccggcg ccacaaactt ttctctgctg aagcaggcag gcgatgtgga ggagaaccct 60
ggacca 66
<210> 5
<211> 66
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCR 13 P2A
<400> 5
ggaagcggag ccaccaactt ttccctgctg aagcaggccg gcgatgtgga ggagaatcct 60
ggccca 66
<210> 6
<211> 66
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCR 12 P2A
<400> 6
ggatctggag ccaccaactt ctccctgctg aagcaggccg gcgatgtgga ggagaatcct 60
ggccca 66
<210> 7
<211> 930
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 14 - бета Кодон-оптимизированная/цистеин-модифицированная
<400> 7
atgggcatcc ggctgctgtg cagagtggcc ttctgttttc tggccgtggg cctggtggac 60
gtgaaggtga cccagagctc ccggtatctg gtgaagagaa caggcgagaa ggtgtttctg 120
gagtgcgtgc aggacatgga tcacgagaac atgttctggt acaggcagga tccaggcctg 180
ggcctgagac tgatctattt cagctacgat gtgaagatga aggagaaggg cgacatccct 240
gagggctatt ctgtgagcag ggagaagaag gagcggttca gcctgatcct ggagtccgcc 300
tctaccaacc agacatctat gtacctgtgc gcaagcacct tctggggaca gaggagaaca 360
gaggccttct ttggccaggg caccaggctg acagtggtgg aggacctgaa taaggtgttc 420
ccccctgagg tggccgtgtt tgagccatcc gaggccgaga tctctcacac ccagaaggcc 480
accctggtgt gcctggcaac cggcttcttt cccgatcacg tggagctgtc ctggtgggtg 540
aacggcaagg aggtgcactc tggcgtgtgc acagacccac agcccctgaa ggagcagcct 600
gccctgaatg atagccgcta ttgtctgtct agcaggctgc gcgtgtccgc caccttttgg 660
cagaacccaa ggaatcactt ccgctgccag gtgcagtttt acggcctgtc cgagaatgac 720
gagtggaccc aggatagggc caagccagtg acacagatcg tgtctgccga ggcatggggc 780
agagccgact gtggcttcac cagcgtgtcc taccagcagg gcgtgctgag cgccaccatc 840
ctgtatgaga tcctgctggg caaggccaca ctgtacgccg tgctggtgtc cgccctggtg 900
ctgatggcca tggtgaagcg gaaggacttc 930
<210> 8
<211> 936
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 13 - бета Кодон-оптимизированная/цистеин-модифицированная
<400> 8
atgggaacca ggctgctgtg ctgggtggtg ctgggctttc tgggaaccga ccacacagga 60
gcaggcgtgt cccagtctcc aaggtacaag gtggccaaga gaggccagga tgtggccctg 120
agatgtgacc ccatctccgg ccacgtgtct ctgttctggt accagcaggc cctgggacag 180
ggaccagagt tcctgacata ttttcagaac gaggcccagc tggataagag cggcctgcct 240
tccgacaggt tctttgcaga gcgcccagag ggaagcgtgt ccaccctgaa gatccagagg 300
acacagcagg aggactccgc cgtgtacctg tgcgcaagct cccctaccgg aacagagagg 360
gagctgttct ttggagaggg cagccgcctg accgtgctgg aggatctgaa gaacgtgttc 420
ccccctgagg tggccgtgtt tgagcctagc gaggccgaga tctcccacac ccagaaggcc 480
accctggtgt gcctggcaac cggcttctat ccagaccacg tggagctgag ctggtgggtg 540
aacggcaagg aggtgcactc cggcgtgtgc acagacccac agcccctgaa ggagcagccc 600
gccctgaatg atagccgcta ctgtctgtct agccggctga gagtgtccgc caccttttgg 660
cagaacccta ggaatcactt ccgctgccag gtgcagtttt atggcctgtc cgagaacgac 720
gagtggaccc aggatcgggc caagcccgtg acacagatcg tgtctgccga ggcatggggc 780
agagccgatt gtggcttcac atctgagagc taccagcagg gcgtgctgtc cgccaccatc 840
ctgtacgaga tcctgctggg caaggccaca ctgtatgccg tgctggtgag cgccctggtg 900
ctgatggcca tggtgaagag gaaggactct agagga 936
<210> 9
<211> 933
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 12 - бета Кодон-оптимизированная/цистеин-модифицированная
<400> 9
atggacacct ggctggtgtg ctgggccatc ttcagcctgc tgaaggcagg cctgaccgag 60
cctgaggtga cccagacacc atcccaccag gtgacacaga tgggccagga agtgatcctg 120
cggtgcgtgc ctatctccaa ccacctgtac ttttattggt acagacagat cctgggccag 180
aaggtggagt ttctggtgag cttctacaac aatgagatca gcgagaagtc cgagatcttt 240
gacgatcagt tctctgtgga gaggcccgac ggcagcaact tcaccctgaa gatccgctcc 300
acaaagctgg aggattctgc catgtatttc tgcgccagca ccacacggag ctcctacgag 360
cagtattttg gccctggcac cagactgacc gtgacagagg acctgaagaa cgtgttcccc 420
cctgaggtgg ccgtgttcga gccatctgag gccgagatca gccacaccca gaaggccacc 480
ctggtgtgcc tggcaaccgg cttctacccc gatcacgtgg agctgagctg gtgggtgaac 540
ggcaaggagg tgcactccgg cgtgtgcaca gacccacagc ccctgaagga gcagcctgcc 600
ctgaatgata gcagatactg tctgtctagc cggctgagag tgtccgccac cttctggcag 660
aacccaagga atcactttcg ctgccaggtg cagttctatg gcctgtctga gaacgacgag 720
tggacccagg atagggccaa gccagtgaca cagatcgtga gcgccgaggc atggggcaga 780
gccgattgtg gctttacaag cgagtcctat cagcagggcg tgctgtccgc caccatcctg 840
tacgagatcc tgctgggcaa ggccacactg tatgccgtgc tggtgtctgc cctggtgctg 900
atggccatgg tgaagaggaa ggactccaga gga 933
<210> 10
<211> 825
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 14 - альфа Кодон-оптимизированная/цистеин-модифицированная
<400> 10
atggagaaga atcctctggc cgccccactg ctgatcctgt ggttccacct ggactgcgtg 60
tcctctatcc tgaatgtgga gcagagccca cagtccctgc acgtgcagga gggcgatagc 120
accaacttca catgttcctt tcctagctcc aacttctacg ccctgcactg gtaccggtgg 180
gagacagcca agagcccaga ggccctgttc gtgatgacac tgaacggcga cgagaagaag 240
aagggcagaa tcagcgccac cctgaataca aaggagggct actcctatct gtacatcaag 300
ggcagccagc ccgaggattc cgccacctac ctgtgcgcct cccagacagg cgccaacaat 360
ctgttctttg gcaccggcac aaggctgacc gtgatccctt atatccagaa cccagaccct 420
gccgtgtacc agctgaggga ctctaagtct agcgataaga gcgtgtgcct gttcaccgac 480
tttgattctc agacaaacgt gagccagagc aaggacagcg acgtgtacat caccgacaag 540
tgcgtgctgg atatgagaag catggacttt aagtccaact ctgccgtggc ctggtctaat 600
aagagcgatt tcgcctgcgc caacgccttt aacaattcca tcatccccga ggatacattc 660
tttccatctc ccgagtcctc ttgtgacgtg aagctggtgg agaagagctt cgagacagat 720
acaaacctga attttcagaa cctgagcgtg atcggcttcc ggatcctgct gctgaaggtg 780
gccggcttca atctgctgat gaccctgaga ctgtggagct cctga 825
<210> 11
<211> 813
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 13 - альфа Кодон-оптимизированная/цистеин-модифицированная
<400> 11
atgctgctgc tgctggtgcc agtgctggaa gtgatcttca ccctgggagg aacaagggca 60
cagtctgtga cccagctgga cagccacgtg tccgtgtctg agggcacacc cgtgctgctg 120
agatgcaact actcctctag ctatagcccc tccctgtttt ggtacgtgca gcaccctaat 180
aagggcctgc agctgctgct gaagtatacc tccgccgcca cactggtgaa gggcatcaat 240
ggcttcgagg ccgagtttaa gaagagcgag acaagcttcc acctgacaaa gccttccgcc 300
cacatgtctg acgccgccga gtacttttgc gtggtgcggg gaggcaagct gatcttcgga 360
cagggaaccg agctgagcgt gaagccaaac atccagaatc ccgatcctgc cgtgtatcag 420
ctgcgcgact ccaagtcctc tgataagagc gtgtgcctgt tcaccgactt tgattctcag 480
acaaacgtgt ctcagagcaa ggacagcgac gtgtacatca ccgacaagtg cgtgctggat 540
atgcggagca tggactttaa gtccaactct gccgtggcct ggtctaataa gagcgatttc 600
gcctgcgcca atgcctttaa caattccatc atccccgagg atacattctt tccatctccc 660
gagagctcct gtgacgtgaa gctggtggag aagagcttcg agacagatac aaacctgaat 720
tttcagaacc tgagcgtgat cggcttcagg atcctgctgc tgaaggtggc cggcttcaat 780
ctgctgatga ccctgcgcct gtggtctagc tga 813
<210> 12
<211> 822
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 12 - альфа
Кодон-оптимизированная/цистеин-модифицированная
<400> 12
atgaagacat ttgccggctt ctcttttctg ttcctgtggc tgcagctgga ttgcatgagc 60
aggggcgagg acgtggagca gagcctgttc ctgtccgtgc gcgagggcga ttcctctgtg 120
atcaactgta cctacacaga cagctcctct acctatctgt actggtataa gcaggagcca 180
ggagcaggcc tgcagctgct gacctatatc ttttccaaca tggacatgaa gcaggatcag 240
cggctgacag tgctgctgaa taagaaggac aagcacctga gcctgagaat cgctgacacc 300
cagacaggcg attccgccat ctacttctgc gccgtgccct ctggcgccac caataagctg 360
atctttggaa ccggcacact gctggcagtg cagcctaaca tccagaatcc cgatcctgcc 420
gtgtaccagc tgcgggacag caagagctcc gataagtccg tgtgcctgtt taccgacttc 480
gattctcaga caaacgtgtc tcagagcaag gacagcgacg tgtacatcac cgacaagtgc 540
gtgctggata tgcggagcat ggacttcaag tccaactctg ccgtggcctg gtctaataag 600
agcgactttg cctgcgccaa tgccttcaac aattccatca tccccgagga tacattcttt 660
ccatctcccg agtctagctg tgacgtgaag ctggtggaga agagcttcga gacagataca 720
aacctgaatt tccagaacct gtctgtgatc ggctttagga tcctgctgct gaaggtggcc 780
ggctttaatc tgctgatgac cctgcgcctg tggtcctctt ga 822
<210> 13
<211> 1821
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 14 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 13
atgggcatcc ggctgctgtg cagagtggcc ttctgttttc tggccgtggg cctggtggac 60
gtgaaggtga cccagagctc ccggtatctg gtgaagagaa caggcgagaa ggtgtttctg 120
gagtgcgtgc aggacatgga tcacgagaac atgttctggt acaggcagga tccaggcctg 180
ggcctgagac tgatctattt cagctacgat gtgaagatga aggagaaggg cgacatccct 240
gagggctatt ctgtgagcag ggagaagaag gagcggttca gcctgatcct ggagtccgcc 300
tctaccaacc agacatctat gtacctgtgc gcaagcacct tctggggaca gaggagaaca 360
gaggccttct ttggccaggg caccaggctg acagtggtgg aggacctgaa taaggtgttc 420
ccccctgagg tggccgtgtt tgagccatcc gaggccgaga tctctcacac ccagaaggcc 480
accctggtgt gcctggcaac cggcttcttt cccgatcacg tggagctgtc ctggtgggtg 540
aacggcaagg aggtgcactc tggcgtgtgc acagacccac agcccctgaa ggagcagcct 600
gccctgaatg atagccgcta ttgtctgtct agcaggctgc gcgtgtccgc caccttttgg 660
cagaacccaa ggaatcactt ccgctgccag gtgcagtttt acggcctgtc cgagaatgac 720
gagtggaccc aggatagggc caagccagtg acacagatcg tgtctgccga ggcatggggc 780
agagccgact gtggcttcac cagcgtgtcc taccagcagg gcgtgctgag cgccaccatc 840
ctgtatgaga tcctgctggg caaggccaca ctgtacgccg tgctggtgtc cgccctggtg 900
ctgatggcca tggtgaagcg gaaggacttc ggctccggcg ccacaaactt ttctctgctg 960
aagcaggcag gcgatgtgga ggagaaccct ggaccaatgg agaagaatcc tctggccgcc 1020
ccactgctga tcctgtggtt ccacctggac tgcgtgtcct ctatcctgaa tgtggagcag 1080
agcccacagt ccctgcacgt gcaggagggc gatagcacca acttcacatg ttcctttcct 1140
agctccaact tctacgccct gcactggtac cggtgggaga cagccaagag cccagaggcc 1200
ctgttcgtga tgacactgaa cggcgacgag aagaagaagg gcagaatcag cgccaccctg 1260
aatacaaagg agggctactc ctatctgtac atcaagggca gccagcccga ggattccgcc 1320
acctacctgt gcgcctccca gacaggcgcc aacaatctgt tctttggcac cggcacaagg 1380
ctgaccgtga tcccttatat ccagaaccca gaccctgccg tgtaccagct gagggactct 1440
aagtctagcg ataagagcgt gtgcctgttc accgactttg attctcagac aaacgtgagc 1500
cagagcaagg acagcgacgt gtacatcacc gacaagtgcg tgctggatat gagaagcatg 1560
gactttaagt ccaactctgc cgtggcctgg tctaataaga gcgatttcgc ctgcgccaac 1620
gcctttaaca attccatcat ccccgaggat acattctttc catctcccga gtcctcttgt 1680
gacgtgaagc tggtggagaa gagcttcgag acagatacaa acctgaattt tcagaacctg 1740
agcgtgatcg gcttccggat cctgctgctg aaggtggccg gcttcaatct gctgatgacc 1800
ctgagactgt ggagctcctg a 1821
<210> 14
<211> 1815
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 13 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 14
atgggaacca ggctgctgtg ctgggtggtg ctgggctttc tgggaaccga ccacacagga 60
gcaggcgtgt cccagtctcc aaggtacaag gtggccaaga gaggccagga tgtggccctg 120
agatgtgacc ccatctccgg ccacgtgtct ctgttctggt accagcaggc cctgggacag 180
ggaccagagt tcctgacata ttttcagaac gaggcccagc tggataagag cggcctgcct 240
tccgacaggt tctttgcaga gcgcccagag ggaagcgtgt ccaccctgaa gatccagagg 300
acacagcagg aggactccgc cgtgtacctg tgcgcaagct cccctaccgg aacagagagg 360
gagctgttct ttggagaggg cagccgcctg accgtgctgg aggatctgaa gaacgtgttc 420
ccccctgagg tggccgtgtt tgagcctagc gaggccgaga tctcccacac ccagaaggcc 480
accctggtgt gcctggcaac cggcttctat ccagaccacg tggagctgag ctggtgggtg 540
aacggcaagg aggtgcactc cggcgtgtgc acagacccac agcccctgaa ggagcagccc 600
gccctgaatg atagccgcta ctgtctgtct agccggctga gagtgtccgc caccttttgg 660
cagaacccta ggaatcactt ccgctgccag gtgcagtttt atggcctgtc cgagaacgac 720
gagtggaccc aggatcgggc caagcccgtg acacagatcg tgtctgccga ggcatggggc 780
agagccgatt gtggcttcac atctgagagc taccagcagg gcgtgctgtc cgccaccatc 840
ctgtacgaga tcctgctggg caaggccaca ctgtatgccg tgctggtgag cgccctggtg 900
ctgatggcca tggtgaagag gaaggactct agaggaggaa gcggagccac caacttttcc 960
ctgctgaagc aggccggcga tgtggaggag aatcctggcc caatgctgct gctgctggtg 1020
ccagtgctgg aagtgatctt caccctggga ggaacaaggg cacagtctgt gacccagctg 1080
gacagccacg tgtccgtgtc tgagggcaca cccgtgctgc tgagatgcaa ctactcctct 1140
agctatagcc cctccctgtt ttggtacgtg cagcacccta ataagggcct gcagctgctg 1200
ctgaagtata cctccgccgc cacactggtg aagggcatca atggcttcga ggccgagttt 1260
aagaagagcg agacaagctt ccacctgaca aagccttccg cccacatgtc tgacgccgcc 1320
gagtactttt gcgtggtgcg gggaggcaag ctgatcttcg gacagggaac cgagctgagc 1380
gtgaagccaa acatccagaa tcccgatcct gccgtgtatc agctgcgcga ctccaagtcc 1440
tctgataaga gcgtgtgcct gttcaccgac tttgattctc agacaaacgt gtctcagagc 1500
aaggacagcg acgtgtacat caccgacaag tgcgtgctgg atatgcggag catggacttt 1560
aagtccaact ctgccgtggc ctggtctaat aagagcgatt tcgcctgcgc caatgccttt 1620
aacaattcca tcatccccga ggatacattc tttccatctc ccgagagctc ctgtgacgtg 1680
aagctggtgg agaagagctt cgagacagat acaaacctga attttcagaa cctgagcgtg 1740
atcggcttca ggatcctgct gctgaaggtg gccggcttca atctgctgat gaccctgcgc 1800
ctgtggtcta gctga 1815
<210> 15
<211> 1821
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 12 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 15
atggacacct ggctggtgtg ctgggccatc ttcagcctgc tgaaggcagg cctgaccgag 60
cctgaggtga cccagacacc atcccaccag gtgacacaga tgggccagga agtgatcctg 120
cggtgcgtgc ctatctccaa ccacctgtac ttttattggt acagacagat cctgggccag 180
aaggtggagt ttctggtgag cttctacaac aatgagatca gcgagaagtc cgagatcttt 240
gacgatcagt tctctgtgga gaggcccgac ggcagcaact tcaccctgaa gatccgctcc 300
acaaagctgg aggattctgc catgtatttc tgcgccagca ccacacggag ctcctacgag 360
cagtattttg gccctggcac cagactgacc gtgacagagg acctgaagaa cgtgttcccc 420
cctgaggtgg ccgtgttcga gccatctgag gccgagatca gccacaccca gaaggccacc 480
ctggtgtgcc tggcaaccgg cttctacccc gatcacgtgg agctgagctg gtgggtgaac 540
ggcaaggagg tgcactccgg cgtgtgcaca gacccacagc ccctgaagga gcagcctgcc 600
ctgaatgata gcagatactg tctgtctagc cggctgagag tgtccgccac cttctggcag 660
aacccaagga atcactttcg ctgccaggtg cagttctatg gcctgtctga gaacgacgag 720
tggacccagg atagggccaa gccagtgaca cagatcgtga gcgccgaggc atggggcaga 780
gccgattgtg gctttacaag cgagtcctat cagcagggcg tgctgtccgc caccatcctg 840
tacgagatcc tgctgggcaa ggccacactg tatgccgtgc tggtgtctgc cctggtgctg 900
atggccatgg tgaagaggaa ggactccaga ggaggatctg gagccaccaa cttctccctg 960
ctgaagcagg ccggcgatgt ggaggagaat cctggcccaa tgaagacatt tgccggcttc 1020
tcttttctgt tcctgtggct gcagctggat tgcatgagca ggggcgagga cgtggagcag 1080
agcctgttcc tgtccgtgcg cgagggcgat tcctctgtga tcaactgtac ctacacagac 1140
agctcctcta cctatctgta ctggtataag caggagccag gagcaggcct gcagctgctg 1200
acctatatct tttccaacat ggacatgaag caggatcagc ggctgacagt gctgctgaat 1260
aagaaggaca agcacctgag cctgagaatc gctgacaccc agacaggcga ttccgccatc 1320
tacttctgcg ccgtgccctc tggcgccacc aataagctga tctttggaac cggcacactg 1380
ctggcagtgc agcctaacat ccagaatccc gatcctgccg tgtaccagct gcgggacagc 1440
aagagctccg ataagtccgt gtgcctgttt accgacttcg attctcagac aaacgtgtct 1500
cagagcaagg acagcgacgt gtacatcacc gacaagtgcg tgctggatat gcggagcatg 1560
gacttcaagt ccaactctgc cgtggcctgg tctaataaga gcgactttgc ctgcgccaat 1620
gccttcaaca attccatcat ccccgaggat acattctttc catctcccga gtctagctgt 1680
gacgtgaagc tggtggagaa gagcttcgag acagatacaa acctgaattt ccagaacctg 1740
tctgtgatcg gctttaggat cctgctgctg aaggtggccg gctttaatct gctgatgacc 1800
ctgcgcctgt ggtcctcttg a 1821
<210> 16
<211> 933
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 14 - бета нативный
<400> 16
atgggaatca ggctcctctg tcgtgtggcc ttttgtttcc tggctgtagg cctcgtagat 60
gtgaaagtaa cccagagctc gagatatcta gtcaaaagga cgggagagaa agtttttctg 120
gaatgtgtcc aggatatgga ccatgaaaat atgttctggt atcgacaaga cccaggtctg 180
gggctacggc tgatctattt ctcatatgat gttaaaatga aagaaaaagg agatattcct 240
gaggggtaca gtgtctctag agagaagaag gagcgcttct ccctgattct ggagtccgcc 300
agcaccaacc agacatctat gtacctctgt gccagcacct tctggggaca gcgaaggact 360
gaagctttct ttggacaagg caccagactc acagttgtag aggacctgaa caaggtgttc 420
ccacccgagg tcgctgtgtt tgagccatca gaagcagaga tctcccacac ccaaaaggcc 480
acactggtgt gcctggccac aggcttcttc cctgaccacg tggagctgag ctggtgggtg 540
aatgggaagg aggtgcacag tggggtcagc acggacccgc agcccctcaa ggagcagccc 600
gccctcaatg actccagata ctgcctgagc agccgcctga gggtctcggc caccttctgg 660
cagaaccccc gcaaccactt ccgctgtcaa gtccagttct acgggctctc ggagaatgac 720
gagtggaccc aggatagggc caaacccgtc acccagatcg tcagcgccga ggcctggggt 780
agagcagact gtggctttac ctcggtgtcc taccagcaag gggtcctgtc tgccaccatc 840
ctctatgaga tcctgctagg gaaggccacc ctgtatgctg tgctggtcag cgcccttgtg 900
ttgatggcca tggtcaagag aaaggatttc tga 933
<210> 17
<211> 939
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 13 - бета нативный
<400> 17
atgggcacca ggctcctctg ctgggtggtc ctgggtttcc tagggacaga tcacacaggt 60
gctggagtct cccagtcccc taggtacaaa gtcgcaaaga gaggacagga tgtagctctc 120
aggtgtgatc caatttcggg tcatgtatcc cttttttggt accaacaggc cctggggcag 180
gggccagagt ttctgactta tttccagaat gaagctcaac tagacaaatc ggggctgccc 240
agtgatcgct tctttgcaga aaggcctgag ggatccgtct ccactctgaa gatccagcgc 300
acacagcagg aggactccgc cgtgtatctc tgtgccagca gcccgacagg gactgagagg 360
gagctgtttt ttggagaagg ctctaggctg accgtactgg aggacctgaa aaacgtgttc 420
ccacccgagg tcgctgtgtt tgagccatca gaagcagaga tctcccacac ccaaaaggcc 480
acactggtgt gcctggccac aggcttctac cccgaccacg tggagctgag ctggtgggtg 540
aatgggaagg aggtgcacag tggggtcagc acagacccgc agcccctcaa ggagcagccc 600
gccctcaatg actccagata ctgcctgagc agccgcctga gggtctcggc caccttctgg 660
cagaaccccc gcaaccactt ccgctgtcaa gtccagttct acgggctctc ggagaatgac 720
gagtggaccc aggatagggc caaacctgtc acccagatcg tcagcgccga ggcctggggt 780
agagcagact gtggcttcac ctccgagtct taccagcaag gggtcctgtc tgccaccatc 840
ctctatgaga tcttgctagg gaaggccacc ttgtatgccg tgctggtcag tgccctcgtg 900
ctgatggcca tggtcaagag aaaggattcc agaggctag 939
<210> 18
<211> 255
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 3 - альфа нативный
<400> 18
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
1 5 10 15
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln Ser Tyr
20 25 30
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
35 40 45
Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly Arg Tyr
50 55 60
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
65 70 75 80
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Arg Glu
85 90 95
Gly Arg Gly Phe Lys Thr Ile Phe Gly Ala Gly Thr Arg Leu Phe Val
100 105 110
Lys Ala Asn Ile Gln Lys Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp
115 120 125
Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser
130 135 140
Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp
145 150 155 160
Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala
165 170 175
Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn
180 185 190
Asn Ser Ile Ile Pro Ala Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser
195 200 205
Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu
210 215 220
Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys
225 230 235 240
Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250 255
<210> 19
<211> 255
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 3 - альфа, цистеин-модифицированный
<400> 19
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
1 5 10 15
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln Ser Tyr
20 25 30
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
35 40 45
Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly Arg Tyr
50 55 60
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
65 70 75 80
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Arg Glu
85 90 95
Gly Arg Gly Phe Lys Thr Ile Phe Gly Ala Gly Thr Arg Leu Phe Val
100 105 110
Lys Ala Asn Ile Gln Lys Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp
115 120 125
Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser
130 135 140
Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp
145 150 155 160
Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala
165 170 175
Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn
180 185 190
Asn Ser Ile Ile Pro Ala Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser
195 200 205
Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu
210 215 220
Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys
225 230 235 240
Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250 255
<210> 20
<211> 816
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 3 - альфа нативный
<400> 20
atgtcacttt ctagcctgct gaaggtggtc acagcttcac tgtggctagg acctggcatt 60
gcccagaaga taactcaaac ccaaccagga atgttcgtgc aggaaaagga ggctgtgact 120
ctggactgca catatgacac cagtgatcaa agttatggtc tcttctggta caagcagccc 180
agcagtgggg aaatgatttt tcttatttat caggggtctt atgacgagca aaatgcaaca 240
gaaggtcgct actcattgaa tttccagaag gcaagaaaat ccgccaacct tgtcatctcc 300
gcttcacaac tgggggactc agcaatgtat ttctgtgcaa tgagagaggg gcgaggcttc 360
aaaactatct ttggagcagg aacaagacta tttgttaaag caaatatcca gaagcctgac 420
cctgccgtgt accagctgag agactctaaa tccagtgaca agtctgtctg cctattcacc 480
gattttgatt ctcaaacaaa tgtgtcacaa agtaaggatt ctgatgtgta tatcacagac 540
aaaactgtgc tagacatgag gtctatggac ttcaagagca acagtgctgt ggcctggagc 600
aacaaatctg actttgcatg tgcaaacgcc ttcaacaaca gcattattcc agcagacacc 660
ttcttcccca gcccagaaag ttcctgtgat gtcaagctgg tcgagaaaag ctttgaaaca 720
gatacgaacc taaactttca aaacctgtca gtgattgggt tccgaatcct cctcctgaaa 780
gtggccgggt ttaatctgct catgacgctg cggctg 816
<210> 21
<211> 825
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 3 - альфа
Кодон-оптимизированный/цистеин-модифицированный
<400> 21
atgtcacttt ctagcctgct gaaggtggtc acagcttcac tgtggctagg acctggcatt 60
gcccagaaga taactcaaac ccaaccagga atgttcgtgc aggaaaagga ggctgtgact 120
ctggactgca catatgacac cagtgatcaa agttatggtc tcttctggta caagcagccc 180
agcagtgggg aaatgatttt tcttatttat caggggtctt atgacgagca aaatgcaaca 240
gaaggtcgct actcattgaa tttccagaag gcaagaaaat ccgccaacct tgtcatctcc 300
gcttcacaac tgggggactc agcaatgtat ttctgtgcaa tgagagaggg gcgaggcttc 360
aaaactatct ttggagcagg aacaagacta tttgttaaag caaatatcca gaagcctgac 420
cctgccgtgt accagctgag agactctaaa tccagtgaca agtctgtctg cctattcacc 480
gattttgatt ctcaaacaaa tgtgtcacaa agtaaggatt ctgatgtgta tatcacagac 540
aaatgtgtgc tagacatgag gtctatggac ttcaagagca acagtgctgt ggcctggagc 600
aacaaatctg actttgcatg tgcaaacgcc ttcaacaaca gcattattcc agcagacacc 660
ttcttcccca gcccagaaag ttcctgtgat gtcaagctgg tcgagaaaag ctttgaaaca 720
gatacgaacc taaactttca aaacctgtca gtgattgggt tccgaatcct cctcctgaaa 780
gtggccgggt ttaatctgct catgacgctg cggctgtggt cttcc 825
<210> 22
<211> 294
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 3 - бета нативный
<400> 22
Gly Ala Gly Val Ser Gln Ser Pro Arg Tyr Lys Val Ala Lys Arg Gly
1 5 10 15
Gln Asp Val Ala Leu Arg Cys Asp Pro Ile Ser Gly His Val Ser Leu
20 25 30
Phe Trp Tyr Gln Gln Ala Leu Gly Gln Gly Pro Glu Phe Leu Thr Tyr
35 40 45
Phe Gln Asn Glu Ala Gln Leu Asp Lys Ser Gly Leu Pro Ser Asp Arg
50 55 60
Phe Phe Ala Glu Arg Pro Glu Gly Ser Val Ser Thr Leu Lys Ile Gln
65 70 75 80
Arg Thr Gln Gln Glu Asp Ser Ala Val Tyr Leu Cys Ala Ser Ser His
85 90 95
Leu Ala Gly Phe Thr Gly Glu Leu Phe Phe Gly Glu Gly Ser Arg Leu
100 105 110
Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val
115 120 125
Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu
130 135 140
Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp
145 150 155 160
Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln
165 170 175
Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser
180 185 190
Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His
195 200 205
Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp
210 215 220
Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala
225 230 235 240
Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly
245 250 255
Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr
260 265 270
Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys
275 280 285
Arg Lys Asp Ser Arg Gly
290
<210> 23
<211> 294
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 3 - бета, цистеин-модифицированный
<400> 23
Gly Ala Gly Val Ser Gln Ser Pro Arg Tyr Lys Val Ala Lys Arg Gly
1 5 10 15
Gln Asp Val Ala Leu Arg Cys Asp Pro Ile Ser Gly His Val Ser Leu
20 25 30
Phe Trp Tyr Gln Gln Ala Leu Gly Gln Gly Pro Glu Phe Leu Thr Tyr
35 40 45
Phe Gln Asn Glu Ala Gln Leu Asp Lys Ser Gly Leu Pro Ser Asp Arg
50 55 60
Phe Phe Ala Glu Arg Pro Glu Gly Ser Val Ser Thr Leu Lys Ile Gln
65 70 75 80
Arg Thr Gln Gln Glu Asp Ser Ala Val Tyr Leu Cys Ala Ser Ser His
85 90 95
Leu Ala Gly Phe Thr Gly Glu Leu Phe Phe Gly Glu Gly Ser Arg Leu
100 105 110
Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val
115 120 125
Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu
130 135 140
Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp
145 150 155 160
Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln
165 170 175
Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser
180 185 190
Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His
195 200 205
Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp
210 215 220
Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala
225 230 235 240
Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly
245 250 255
Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr
260 265 270
Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys
275 280 285
Arg Lys Asp Ser Arg Gly
290
<210> 24
<211> 939
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 3 - бета нативный
<400> 24
atgggcacca ggctcctctg ctgggtggtc ctgggtttcc tagggacaga tcacacaggt 60
gctggagtct cccagtcccc taggtacaaa gtcgcaaaga gaggacagga tgtagctctc 120
aggtgtgatc caatttcggg tcatgtatcc cttttttggt accaacaggc cctggggcag 180
gggccagagt ttctgactta tttccagaat gaagctcaac tagacaaatc ggggctgccc 240
agtgatcgct tctttgcaga aaggcctgag ggatccgtct ccactctgaa gatccagcgc 300
acacagcagg aggactccgc cgtgtatctc tgtgccagca gccacctcgc cgggttcacc 360
ggggagctgt tttttggaga aggctctagg ctgaccgtac tggaggacct gaaaaacgtg 420
ttcccacccg aggtcgctgt gtttgagcca tcagaagcag agatctccca cacccaaaag 480
gccacactgg tgtgcctggc cacaggcttc taccccgacc acgtggagct gagctggtgg 540
gtgaatggga aggaggtgca cagtggggtc agcacagacc cgcagcccct caaggagcag 600
cccgccctca atgactccag atactgcctg agcagccgcc tgagggtctc ggccaccttc 660
tggcagaacc cccgcaacca cttccgctgt caagtccagt tctacgggct ctcggagaat 720
gacgagtgga cccaggatag ggccaaacct gtcacccaga tcgtcagcgc cgaggcctgg 780
ggtagagcag actgtggctt cacctccgag tcttaccagc aaggggtcct gtctgccacc 840
atcctctatg agatcttgct agggaaggcc accttgtatg ccgtgctggt cagtgccctc 900
gtgctgatgg ccatggtcaa gagaaaggat tccagaggc 939
<210> 25
<211> 939
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 3 - бета, Кодон-оптимизированный/цистеин-модифицированный
<400> 25
atgggcacca ggctcctctg ctgggtggtc ctgggtttcc tagggacaga tcacacaggt 60
gctggagtct cccagtcccc taggtacaaa gtcgcaaaga gaggacagga tgtagctctc 120
aggtgtgatc caatttcggg tcatgtatcc cttttttggt accaacaggc cctggggcag 180
gggccagagt ttctgactta tttccagaat gaagctcaac tagacaaatc ggggctgccc 240
agtgatcgct tctttgcaga aaggcctgag ggatccgtct ccactctgaa gatccagcgc 300
acacagcagg aggactccgc cgtgtatctc tgtgccagca gccacctcgc cgggttcacc 360
ggggagctgt tttttggaga aggctctagg ctgaccgtac tggaggacct gaaaaacgtg 420
ttcccacccg aggtcgctgt gtttgagcca tcagaagcag agatctccca cacccaaaag 480
gccacactgg tgtgcctggc cacaggcttc taccccgacc acgtggagct gagctggtgg 540
gtgaatggga aggaggtgca cagtggggtc tgtacagacc cgcagcccct caaggagcag 600
cccgccctca atgactccag atactgcctg agcagccgcc tgagggtctc ggccaccttc 660
tggcagaacc cccgcaacca cttccgctgt caagtccagt tctacgggct ctcggagaat 720
gacgagtgga cccaggatag ggccaaacct gtcacccaga tcgtcagcgc cgaggcctgg 780
ggtagagcag actgtggctt cacctccgag tcttaccagc aaggggtcct gtctgccacc 840
atcctctatg agatcttgct agggaaggcc accttgtatg ccgtgctggt cagtgccctc 900
gtgctgatgg ccatggtcaa gagaaaggat tccagaggc 939
<210> 26
<211> 1853
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 3 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 26
gcggccgcca ccatgggcac caggctcctc tgctgggtgg tcctgggttt cctagggaca 60
gatcacacag gtgctggagt ctcccagtcc cctaggtaca aagtcgcaaa gagaggacag 120
gatgtagctc tcaggtgtga tccaatttcg ggtcatgtat cccttttttg gtaccaacag 180
gccctggggc aggggccaga gtttctgact tatttccaga atgaagctca actagacaaa 240
tcggggctgc ccagtgatcg cttctttgca gaaaggcctg agggatccgt ctccactctg 300
aagatccagc gcacacagca ggaggactcc gccgtgtatc tctgtgccag cagccacctc 360
gccgggttca ccggggagct gttttttgga gaaggctcta ggctgaccgt actggaggac 420
ctgaaaaacg tgttcccacc cgaggtcgct gtgtttgagc catcagaagc agagatctcc 480
cacacccaaa aggccacact ggtgtgcctg gccacaggct tctaccccga ccacgtggag 540
ctgagctggt gggtgaatgg gaaggaggtg cacagtgggg tctgtacaga cccgcagccc 600
ctcaaggagc agcccgccct caatgactcc agatactgcc tgagcagccg cctgagggtc 660
tcggccacct tctggcagaa cccccgcaac cacttccgct gtcaagtcca gttctacggg 720
ctctcggaga atgacgagtg gacccaggat agggccaaac ctgtcaccca gatcgtcagc 780
gccgaggcct ggggtagagc agactgtggc ttcacctccg agtcttacca gcaaggggtc 840
ctgtctgcca ccatcctcta tgagatcttg ctagggaagg ccaccttgta tgccgtgctg 900
gtcagtgccc tcgtgctgat ggccatggtc aagagaaagg attccagagg cggatccgga 960
gctaccaact tctctctgct gaaacaggca ggcgatgtgg aggaaaatcc tgggccaatg 1020
tcactttcta gcctgctgaa ggtggtcaca gcttcactgt ggctaggacc tggcattgcc 1080
cagaagataa ctcaaaccca accaggaatg ttcgtgcagg aaaaggaggc tgtgactctg 1140
gactgcacat atgacaccag tgatcaaagt tatggtctct tctggtacaa gcagcccagc 1200
agtggggaaa tgatttttct tatttatcag gggtcttatg acgagcaaaa tgcaacagaa 1260
ggtcgctact cattgaattt ccagaaggca agaaaatccg ccaaccttgt catctccgct 1320
tcacaactgg gggactcagc aatgtatttc tgtgcaatga gagaggggcg aggcttcaaa 1380
actatctttg gagcaggaac aagactattt gttaaagcaa atatccagaa gcctgaccct 1440
gccgtgtacc agctgagaga ctctaaatcc agtgacaagt ctgtctgcct attcaccgat 1500
tttgattctc aaacaaatgt gtcacaaagt aaggattctg atgtgtatat cacagacaaa 1560
tgtgtgctag acatgaggtc tatggacttc aagagcaaca gtgctgtggc ctggagcaac 1620
aaatctgact ttgcatgtgc aaacgccttc aacaacagca ttattccagc agacaccttc 1680
ttccccagcc cagaaagttc ctgtgatgtc aagctggtcg agaaaagctt tgaaacagat 1740
acgaacctaa actttcaaaa cctgtcagtg attgggttcc gaatcctcct cctgaaagtg 1800
gccgggttta atctgctcat gacgctgcgg ctgtggtctt cctaaggcgc gcc 1853
<210> 27
<211> 610
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 3 Полноразмерная последовательность, цистеин-модифицированная
<400> 27
Met Gly Thr Arg Leu Leu Cys Trp Val Val Leu Gly Phe Leu Gly Thr
1 5 10 15
Asp His Thr Gly Ala Gly Val Ser Gln Ser Pro Arg Tyr Lys Val Ala
20 25 30
Lys Arg Gly Gln Asp Val Ala Leu Arg Cys Asp Pro Ile Ser Gly His
35 40 45
Val Ser Leu Phe Trp Tyr Gln Gln Ala Leu Gly Gln Gly Pro Glu Phe
50 55 60
Leu Thr Tyr Phe Gln Asn Glu Ala Gln Leu Asp Lys Ser Gly Leu Pro
65 70 75 80
Ser Asp Arg Phe Phe Ala Glu Arg Pro Glu Gly Ser Val Ser Thr Leu
85 90 95
Lys Ile Gln Arg Thr Gln Gln Glu Asp Ser Ala Val Tyr Leu Cys Ala
100 105 110
Ser Ser His Leu Ala Gly Phe Thr Gly Glu Leu Phe Phe Gly Glu Gly
115 120 125
Ser Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu
130 135 140
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
145 150 155 160
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu
165 170 175
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr
180 185 190
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
195 200 205
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
210 215 220
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
225 230 235 240
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
245 250 255
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr
260 265 270
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
275 280 285
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
290 295 300
Met Val Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe
305 310 315 320
Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met
325 330 335
Ser Leu Ser Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu Gly
340 345 350
Pro Gly Ile Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val
355 360 365
Gln Glu Lys Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp
370 375 380
Gln Ser Tyr Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met
385 390 395 400
Ile Phe Leu Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu
405 410 415
Gly Arg Tyr Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu
420 425 430
Val Ile Ser Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala
435 440 445
Met Arg Glu Gly Arg Gly Phe Lys Thr Ile Phe Gly Ala Gly Thr Arg
450 455 460
Leu Phe Val Lys Ala Asn Ile Gln Lys Pro Asp Pro Ala Val Tyr Gln
465 470 475 480
Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp
485 490 495
Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr
500 505 510
Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser
515 520 525
Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn
530 535 540
Ala Phe Asn Asn Ser Ile Ile Pro Ala Asp Thr Phe Phe Pro Ser Pro
545 550 555 560
Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp
565 570 575
Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu
580 585 590
Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp
595 600 605
Ser Ser
610
<210> 28
<211> 254
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 4 - (E6) 29 альфа нативный
<400> 28
Asp Ala Lys Thr Thr Gln Pro Asn Ser Met Glu Ser Asn Glu Glu Glu
1 5 10 15
Pro Val His Leu Pro Cys Asn His Ser Thr Ile Ser Gly Thr Asp Tyr
20 25 30
Ile His Trp Tyr Arg Gln Leu Pro Ser Gln Gly Pro Glu Tyr Val Ile
35 40 45
His Gly Leu Thr Ser Asn Val Asn Asn Arg Met Ala Ser Leu Ala Ile
50 55 60
Ala Glu Asp Arg Lys Ser Ser Thr Leu Ile Leu His Arg Ala Thr Leu
65 70 75 80
Arg Asp Ala Ala Val Tyr Tyr Cys Ile Leu Leu Val Ile Arg Gly Thr
85 90 95
Ser Tyr Gly Lys Leu Thr Phe Gly Gln Gly Thr Ile Leu Thr Val His
100 105 110
Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser
115 120 125
Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln
130 135 140
Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys
145 150 155 160
Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val
165 170 175
Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn
180 185 190
Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys
195 200 205
Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn
210 215 220
Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val
225 230 235 240
Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 29
<211> 254
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 4 - (E6)29 альфа, цистеин-модифицированный
<400> 29
Asp Ala Lys Thr Thr Gln Pro Asn Ser Met Glu Ser Asn Glu Glu Glu
1 5 10 15
Pro Val His Leu Pro Cys Asn His Ser Thr Ile Ser Gly Thr Asp Tyr
20 25 30
Ile His Trp Tyr Arg Gln Leu Pro Ser Gln Gly Pro Glu Tyr Val Ile
35 40 45
His Gly Leu Thr Ser Asn Val Asn Asn Arg Met Ala Ser Leu Ala Ile
50 55 60
Ala Glu Asp Arg Lys Ser Ser Thr Leu Ile Leu His Arg Ala Thr Leu
65 70 75 80
Arg Asp Ala Ala Val Tyr Tyr Cys Ile Leu Leu Val Ile Arg Gly Thr
85 90 95
Ser Tyr Gly Lys Leu Thr Phe Gly Gln Gly Thr Ile Leu Thr Val His
100 105 110
Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser
115 120 125
Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln
130 135 140
Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys
145 150 155 160
Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val
165 170 175
Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn
180 185 190
Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys
195 200 205
Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn
210 215 220
Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val
225 230 235 240
Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 30
<211> 804
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 4 - (E6)29 альфа нативный
<400> 30
atgaagttgg tgacaagcat tactgtactc ctatctttgg gtattatggg tgatgctaag 60
accacacagc caaattcaat ggagagtaac gaagaagagc ctgttcactt gccttgtaac 120
cactccacaa tcagtggaac tgattacata cattggtatc gacagcttcc ctcccagggt 180
ccagagtacg tgattcatgg tcttacaagc aatgtgaaca acagaatggc ctctctggca 240
atcgctgaag acagaaagtc cagtaccttg atcctgcacc gtgctacctt gagagatgct 300
gctgtgtact actgcatcct actggtaatc cgtggtacta gctatggaaa gctgacattt 360
ggacaaggga ccatcttgac tgtccatcca aatatccaga accctgaccc tgccgtgtac 420
cagctgagag actctaaatc cagtgacaag tctgtctgcc tattcaccga ttttgattct 480
caaacaaatg tgtcacaaag taaggattct gatgtgtata tcacagacaa aactgtgcta 540
gacatgaggt ctatggactt caagagcaac agtgctgtgg cctggagcaa caaatctgac 600
tttgcatgtg caaacgcctt caacaacagc attattccag aagacacctt cttccccagc 660
ccagaaagtt cctgtgatgt caagctggtc gagaaaagct ttgaaacaga tacgaaccta 720
aactttcaaa acctgtcagt gattgggttc cgaatcctcc tcctgaaagt ggccgggttt 780
aatctgctca tgacgctgcg gctg 804
<210> 31
<211> 813
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 4 - (E6)29 альфа кодон-оптимизированный /
цистеин-модифицированный
<400> 31
atgaaactgg tgaccagcat cacagtcctg ctgtccctgg gaattatggg cgacgccaag 60
accacacagc ctaactctat ggagagtaat gaggaagagc ctgtgcacct gccatgtaac 120
cattcaacta tcagcggcac cgattacatt cactggtatc ggcagctgcc ctcccaggga 180
cctgaatacg tgatccatgg cctgacctca aatgtcaaca atcgcatggc tagcctggct 240
atcgcagagg accgaaagtc aagcaccctg attctgcacc gagccacact gcgagatgca 300
gccgtgtact attgcatcct gctggtcatt agagggacca gctacggaaa actgacattt 360
ggccagggga ctatcctgac cgtgcatcct aacattcaga atcccgaccc tgccgtgtat 420
cagctgaggg actctaagtc ctctgataaa agcgtgtgcc tgttcactga ctttgattcc 480
cagaccaacg tgtcccagtc taaggactct gacgtgtaca tcacagacaa atgcgtcctg 540
gatatgcgca gcatggactt caagagtaac tcagccgtgg cttggtccaa caagtctgat 600
ttcgcatgcg ccaacgcttt taacaacagt atcatcccag aagatacctt ctttccatca 660
cccgagagtt catgtgacgt gaagctggtc gaaaaatctt tcgagactga taccaacctg 720
aattttcaga acctgagtgt gatcgggttc aggattctgc tgctgaaggt cgccggattc 780
aatctgctga tgacactgcg cctgtggagc tcc 813
<210> 32
<211> 291
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 4 - (E6)29 бета нативный
<400> 32
Asp Thr Gly Val Ser Gln Asp Pro Arg His Lys Ile Thr Lys Arg Gly
1 5 10 15
Gln Asn Val Thr Phe Arg Cys Asp Pro Ile Ser Glu His Asn Arg Leu
20 25 30
Tyr Trp Tyr Arg Gln Thr Leu Gly Gln Gly Pro Glu Phe Leu Thr Tyr
35 40 45
Phe Gln Asn Glu Ala Gln Leu Glu Lys Ser Arg Leu Leu Ser Asp Arg
50 55 60
Phe Ser Ala Glu Arg Pro Lys Gly Ser Phe Ser Thr Leu Glu Ile Gln
65 70 75 80
Arg Thr Glu Gln Gly Asp Ser Ala Met Tyr Leu Cys Ala Ser Ser Pro
85 90 95
Gly Gly Gly Asn Thr Glu Ala Phe Phe Gly Gln Gly Thr Arg Leu Thr
100 105 110
Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val Phe
115 120 125
Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val
130 135 140
Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp Trp
145 150 155 160
Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro
165 170 175
Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser
180 185 190
Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe
195 200 205
Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr
210 215 220
Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp
225 230 235 240
Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly Val
245 250 255
Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu
260 265 270
Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg
275 280 285
Lys Asp Phe
290
<210> 33
<211> 291
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 4 - (E6)29 бета, цистеин-модифицированный
<400> 33
Asp Thr Gly Val Ser Gln Asp Pro Arg His Lys Ile Thr Lys Arg Gly
1 5 10 15
Gln Asn Val Thr Phe Arg Cys Asp Pro Ile Ser Glu His Asn Arg Leu
20 25 30
Tyr Trp Tyr Arg Gln Thr Leu Gly Gln Gly Pro Glu Phe Leu Thr Tyr
35 40 45
Phe Gln Asn Glu Ala Gln Leu Glu Lys Ser Arg Leu Leu Ser Asp Arg
50 55 60
Phe Ser Ala Glu Arg Pro Lys Gly Ser Phe Ser Thr Leu Glu Ile Gln
65 70 75 80
Arg Thr Glu Gln Gly Asp Ser Ala Met Tyr Leu Cys Ala Ser Ser Pro
85 90 95
Gly Gly Gly Asn Thr Glu Ala Phe Phe Gly Gln Gly Thr Arg Leu Thr
100 105 110
Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val Phe
115 120 125
Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val
130 135 140
Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp Trp
145 150 155 160
Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro
165 170 175
Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser
180 185 190
Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe
195 200 205
Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr
210 215 220
Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp
225 230 235 240
Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly Val
245 250 255
Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu
260 265 270
Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg
275 280 285
Lys Asp Phe
290
<210> 34
<211> 930
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 4 - (E6)29 бета нативный
<400> 34
atgggcacca gcctcctctg ctggatggcc ctgtgtctcc tgggggcaga tcacgcagat 60
actggagtct cccaggaccc cagacacaag atcacaaaga ggggacagaa tgtaactttc 120
aggtgtgatc caatttctga acacaaccgc ctttattggt accgacagac cctggggcag 180
ggcccagagt ttctgactta cttccagaat gaagctcaac tagaaaaatc aaggctgctc 240
agtgatcggt tctctgcaga gaggcctaag ggatctttct ccaccttgga gatccagcgc 300
acagagcagg gggactcggc catgtatctc tgtgccagca gccccggcgg ggggaacact 360
gaagctttct ttggacaagg caccagactc acagttgtag aggacctgaa caaggtgttc 420
ccacccgagg tcgctgtgtt tgagccatca gaagcagaga tctcccacac ccaaaaggcc 480
acactggtgt gcctggccac aggcttcttc cctgaccacg tggagctgag ctggtgggtg 540
aatgggaagg aggtgcacag tggggtcagc acggacccgc agcccctcaa ggagcagccc 600
gccctcaatg actccagata ctgcctgagc agccgcctga gggtctcggc caccttctgg 660
cagaaccccc gcaaccactt ccgctgtcaa gtccagttct acgggctctc ggagaatgac 720
gagtggaccc aggatagggc caaacccgtc acccagatcg tcagcgccga ggcctggggt 780
agagcagact gtggctttac ctcggtgtcc taccagcaag gggtcctgtc tgccaccatc 840
ctctatgaga tcctgctagg gaaggccacc ctgtatgctg tgctggtcag cgcccttgtg 900
ttgatggcca tggtcaagag aaaggatttc 930
<210> 35
<211> 930
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 4 - (E6)29 бета, кодон-оптимизированный /
цистеин-модифицированный
<400> 35
atggggacta gcctgctgtg ctggatggca ctgtgcctgc tgggagcaga ccacgcagat 60
accggagtga gccaggaccc aagacataag atcacaaaaa ggggccagaa cgtgactttt 120
agatgcgatc ccattagcga acacaataga ctgtactggt ataggcagac actgggacag 180
ggaccagagt tcctgactta ctttcagaac gaagctcagc tggagaagag tcgcctgctg 240
tcagaccggt tcagcgccga gcgaccaaaa ggctctttca gtacactgga aatccagcga 300
actgagcagg gggattccgc catgtatctg tgcgctagct ccccaggagg aggaaacacc 360
gaagccttct ttggacaggg cacacggctg actgtggtcg aggacctgaa taaggtgttc 420
ccccctgaag tggccgtctt tgagccttcc gaagctgaga tttctcacac ccagaaagcc 480
accctggtgt gcctggcaac aggcttcttt ccagatcacg tggaactgag ctggtgggtc 540
aacggaaagg aggtgcatag cggcgtctgc actgacccac agcccctgaa agagcagccc 600
gcactgaatg atagcaggta ctgcctgtct agtcggctga gagtgtccgc caccttttgg 660
cagaacccta ggaatcattt ccgctgtcag gtgcagtttt atggcctgtc cgaaaacgac 720
gagtggactc aggatcgggc caagcccgtg acccagatcg tctctgcaga agcctggggc 780
agagctgact gcgggttcac ctcagtgagc taccagcagg gagtcctgtc cgctaccatc 840
ctgtacgaga ttctgctggg caaggctaca ctgtatgcag tgctggtctc tgcactggtg 900
ctgatggcca tggtcaagcg caaagacttc 930
<210> 36
<211> 1832
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 4 - (E6)29 кодон-оптимизированный /
цистеин-модифицированный, полноразмерная последовательность
<400> 36
gcggccgcca ccatggggac tagcctgctg tgctggatgg cactgtgcct gctgggagca 60
gaccacgcag ataccggagt gagccaggac ccaagacata agatcacaaa aaggggccag 120
aacgtgactt ttagatgcga tcccattagc gaacacaata gactgtactg gtataggcag 180
acactgggac agggaccaga gttcctgact tactttcaga acgaagctca gctggagaag 240
agtcgcctgc tgtcagaccg gttcagcgcc gagcgaccaa aaggctcttt cagtacactg 300
gaaatccagc gaactgagca gggggattcc gccatgtatc tgtgcgctag ctccccagga 360
ggaggaaaca ccgaagcctt ctttggacag ggcacacggc tgactgtggt cgaggacctg 420
aataaggtgt tcccccctga agtggccgtc tttgagcctt ccgaagctga gatttctcac 480
acccagaaag ccaccctggt gtgcctggca acaggcttct ttccagatca cgtggaactg 540
agctggtggg tcaacggaaa ggaggtgcat agcggcgtct gcactgaccc acagcccctg 600
aaagagcagc ccgcactgaa tgatagcagg tactgcctgt ctagtcggct gagagtgtcc 660
gccacctttt ggcagaaccc taggaatcat ttccgctgtc aggtgcagtt ttatggcctg 720
tccgaaaacg acgagtggac tcaggatcgg gccaagcccg tgacccagat cgtctctgca 780
gaagcctggg gcagagctga ctgcgggttc acctcagtga gctaccagca gggagtcctg 840
tccgctacca tcctgtacga gattctgctg ggcaaggcta cactgtatgc agtgctggtc 900
tctgcactgg tgctgatggc catggtcaag cgcaaagact tcgggagtgg agcaacaaac 960
ttttcactgc tgaagcaggc cggcgatgtg gaggaaaatc ctgggccaat gaaactggtg 1020
accagcatca cagtcctgct gtccctggga attatgggcg acgccaagac cacacagcct 1080
aactctatgg agagtaatga ggaagagcct gtgcacctgc catgtaacca ttcaactatc 1140
agcggcaccg attacattca ctggtatcgg cagctgccct cccagggacc tgaatacgtg 1200
atccatggcc tgacctcaaa tgtcaacaat cgcatggcta gcctggctat cgcagaggac 1260
cgaaagtcaa gcaccctgat tctgcaccga gccacactgc gagatgcagc cgtgtactat 1320
tgcatcctgc tggtcattag agggaccagc tacggaaaac tgacatttgg ccaggggact 1380
atcctgaccg tgcatcctaa cattcagaat cccgaccctg ccgtgtatca gctgagggac 1440
tctaagtcct ctgataaaag cgtgtgcctg ttcactgact ttgattccca gaccaacgtg 1500
tcccagtcta aggactctga cgtgtacatc acagacaaat gcgtcctgga tatgcgcagc 1560
atggacttca agagtaactc agccgtggct tggtccaaca agtctgattt cgcatgcgcc 1620
aacgctttta acaacagtat catcccagaa gataccttct ttccatcacc cgagagttca 1680
tgtgacgtga agctggtcga aaaatctttc gagactgata ccaacctgaa ttttcagaac 1740
ctgagtgtga tcgggttcag gattctgctg ctgaaggtcg ccggattcaa tctgctgatg 1800
acactgcgcc tgtggagctc ctgaggcgcg cc 1832
<210> 37
<211> 603
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 4 - (E6)29 Полноразмерная последовательность, цистеин-модифицированная
<400> 37
Met Gly Thr Ser Leu Leu Cys Trp Met Ala Leu Cys Leu Leu Gly Ala
1 5 10 15
Asp His Ala Asp Thr Gly Val Ser Gln Asp Pro Arg His Lys Ile Thr
20 25 30
Lys Arg Gly Gln Asn Val Thr Phe Arg Cys Asp Pro Ile Ser Glu His
35 40 45
Asn Arg Leu Tyr Trp Tyr Arg Gln Thr Leu Gly Gln Gly Pro Glu Phe
50 55 60
Leu Thr Tyr Phe Gln Asn Glu Ala Gln Leu Glu Lys Ser Arg Leu Leu
65 70 75 80
Ser Asp Arg Phe Ser Ala Glu Arg Pro Lys Gly Ser Phe Ser Thr Leu
85 90 95
Glu Ile Gln Arg Thr Glu Gln Gly Asp Ser Ala Met Tyr Leu Cys Ala
100 105 110
Ser Ser Pro Gly Gly Gly Asn Thr Glu Ala Phe Phe Gly Gln Gly Thr
115 120 125
Arg Leu Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu
305 310 315 320
Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Lys Leu Val
325 330 335
Thr Ser Ile Thr Val Leu Leu Ser Leu Gly Ile Met Gly Asp Ala Lys
340 345 350
Thr Thr Gln Pro Asn Ser Met Glu Ser Asn Glu Glu Glu Pro Val His
355 360 365
Leu Pro Cys Asn His Ser Thr Ile Ser Gly Thr Asp Tyr Ile His Trp
370 375 380
Tyr Arg Gln Leu Pro Ser Gln Gly Pro Glu Tyr Val Ile His Gly Leu
385 390 395 400
Thr Ser Asn Val Asn Asn Arg Met Ala Ser Leu Ala Ile Ala Glu Asp
405 410 415
Arg Lys Ser Ser Thr Leu Ile Leu His Arg Ala Thr Leu Arg Asp Ala
420 425 430
Ala Val Tyr Tyr Cys Ile Leu Leu Val Ile Arg Gly Thr Ser Tyr Gly
435 440 445
Lys Leu Thr Phe Gly Gln Gly Thr Ile Leu Thr Val His Pro Asn Ile
450 455 460
Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser
465 470 475 480
Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val
485 490 495
Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys Val Leu
500 505 510
Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser
515 520 525
Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile
530 535 540
Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys
545 550 555 560
Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn
565 570 575
Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe
580 585 590
Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600
<210> 38
<211> 258
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 5 - (E6)29 - TCR альфа нативный
<400> 38
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
1 5 10 15
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln Ser Tyr
20 25 30
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
35 40 45
Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly Arg Tyr
50 55 60
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
65 70 75 80
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Arg Glu
85 90 95
Gly Thr Gly Thr Ser Tyr Gly Lys Leu Thr Phe Gly Gln Gly Thr Ile
100 105 110
Leu Thr Val His Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln
115 120 125
Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp
130 135 140
Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr
145 150 155 160
Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser
165 170 175
Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn
180 185 190
Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro
195 200 205
Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp
210 215 220
Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu
225 230 235 240
Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp
245 250 255
Ser Ser
<210> 39
<211> 258
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 5 - (E6)29 - TCR альфа, цистеин-модифицированный
<400> 39
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
1 5 10 15
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln Ser Tyr
20 25 30
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
35 40 45
Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly Arg Tyr
50 55 60
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
65 70 75 80
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Arg Glu
85 90 95
Gly Thr Gly Thr Ser Tyr Gly Lys Leu Thr Phe Gly Gln Gly Thr Ile
100 105 110
Leu Thr Val His Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln
115 120 125
Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp
130 135 140
Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr
145 150 155 160
Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser
165 170 175
Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn
180 185 190
Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro
195 200 205
Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp
210 215 220
Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu
225 230 235 240
Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp
245 250 255
Ser Ser
<210> 40
<211> 825
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 5 - (E6)29 - TCR альфа нативный
<400> 40
atgtcacttt ctagcctgct gaaggtggtc acagcttcac tgtggctagg acctggcatt 60
gcccagaaga taactcaaac ccaaccagga atgttcgtgc aggaaaagga ggctgtgact 120
ctggactgca catatgacac cagtgatcaa agttatggtc tattctggta caagcagccc 180
agcagtgggg aaatgatttt tcttatttat caggggtctt atgacgagca aaatgcaaca 240
gaaggtcgct actcattgaa tttccagaag gcaagaaaat ccgccaacct tgtcatctcc 300
gcttcacaac tgggggactc agcaatgtat ttctgtgcaa tgagagaggg cacaggtact 360
agctatggaa agctgacatt tggacaaggg accatcttga ctgtccatcc aaatatccag 420
aaccctgacc ctgccgtgta ccagctgaga gactctaaat ccagtgacaa gtctgtctgc 480
ctattcaccg attttgattc tcaaacaaat gtgtcacaaa gtaaggattc tgatgtgtat 540
atcacagaca aaactgtgct agacatgagg tctatggact tcaagagcaa cagtgctgtg 600
gcctggagca acaaatctga ctttgcatgt gcaaacgcct tcaacaacag cattattcca 660
gaagacacct tcttccccag cccagaaagt tcctgtgatg tcaagctggt cgagaaaagc 720
tttgaaacag atacgaacct aaactttcaa aacctgtcag tgattgggtt ccgaatcctc 780
ctcctgaaag tggccgggtt taatctgctc atgacgctgc ggctg 825
<210> 41
<211> 834
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 5 - (E6)29 - TCR альфа, кодон-оптимизированный /
цистеин-модифицированный
<400> 41
atgagtctgt cctctctgct gaaggtggtc actgcatcac tgtggctggg accaggaatc 60
gcacagaaaa ttacccagac acagcctggc atgtttgtcc aggagaagga agccgtgacc 120
ctggactgta cttacgacac cagcgatcag tcctacgggc tgttttggta taagcagcca 180
agttcaggag agatgatctt cctgatctac cagggcagct atgacgagca gaacgctaca 240
gaaggcaggt atagcctgaa tttccagaaa gcccgcaagt ccgctaacct ggtcatctct 300
gccagtcagc tgggggattc tgccatgtac ttttgcgcta tgagggaggg aactggcacc 360
agctatggaa agctgacctt cgggcaggga acaatcctga ctgtccatcc caacattcag 420
aatccagacc ctgccgtgta ccagctgcga gacagtaaaa gctccgataa gagcgtgtgc 480
ctgttcacag actttgattc tcagactaac gtgagccaga gcaaagacag tgatgtctat 540
attaccgaca agtgcgtgct ggatatgcgc agcatggact ttaaatccaa ctctgcagtg 600
gcctggtcta ataagagtga tttcgcttgc gcaaacgcct ttaacaattc aatcattccc 660
gaggatacct tctttccaag ccccgaatct agttgtgacg tgaaactggt ggagaagtct 720
ttcgaaacag atactaacct gaattttcag aatctgagtg tcatcgggtt ccggattctg 780
ctgctgaagg tggccggatt caacctgctg atgaccctga gactgtggtc aagc 834
<210> 42
<211> 291
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 5 - (E6)29 - TCR бета нативный
<400> 42
Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys Arg Thr Gly
1 5 10 15
Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His Glu Asn Met
20 25 30
Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu Ile Tyr Phe
35 40 45
Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro Glu Gly Tyr
50 55 60
Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile Leu Glu Ser
65 70 75 80
Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser Ser Pro Trp
85 90 95
Gly Glu Thr His Gln Pro Gln His Phe Gly Asp Gly Thr Arg Leu Ser
100 105 110
Ile Leu Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val Phe
115 120 125
Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val
130 135 140
Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp Trp
145 150 155 160
Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro
165 170 175
Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser
180 185 190
Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe
195 200 205
Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr
210 215 220
Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp
225 230 235 240
Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly Val
245 250 255
Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu
260 265 270
Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg
275 280 285
Lys Asp Phe
290
<210> 43
<211> 291
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 5 - (E6)29 - TCR бета, цистеин-модифицированный
<400> 43
Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys Arg Thr Gly
1 5 10 15
Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His Glu Asn Met
20 25 30
Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu Ile Tyr Phe
35 40 45
Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro Glu Gly Tyr
50 55 60
Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile Leu Glu Ser
65 70 75 80
Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser Ser Pro Trp
85 90 95
Gly Glu Thr His Gln Pro Gln His Phe Gly Asp Gly Thr Arg Leu Ser
100 105 110
Ile Leu Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val Phe
115 120 125
Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val
130 135 140
Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp Trp
145 150 155 160
Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro
165 170 175
Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser
180 185 190
Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe
195 200 205
Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr
210 215 220
Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp
225 230 235 240
Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly Val
245 250 255
Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu
260 265 270
Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg
275 280 285
Lys Asp Phe
290
<210> 44
<211> 930
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 5 - (E6)29 - TCR бета нативный
<400> 44
atgggaatca ggctcctctg tcgtgtggcc ttttgtttcc tggctgtagg cctcgtagat 60
gtgaaagtaa cccagagctc gagatatcta gtcaaaagga cgggagagaa agtttttctg 120
gaatgtgtcc aggatatgga ccatgaaaat atgttctggt atcgacaaga cccaggtctg 180
gggctacggc tgatctattt ctcatatgat gttaaaatga aagaaaaagg agatattcct 240
gaggggtaca gtgtctctag agagaagaag gagcgcttct ccctgattct ggagtccgcc 300
agcaccaacc agacatctat gtacctctgt gccagcagcc catggggaga aactcatcag 360
ccccagcatt ttggtgatgg gactcgactc tccatcctag aggacctgaa caaggtgttc 420
ccacccgagg tcgctgtgtt tgagccatca gaagcagaga tctcccacac ccaaaaggcc 480
acactggtgt gcctggccac aggcttcttc cctgaccacg tggagctgag ctggtgggtg 540
aatgggaagg aggtgcacag tggggtcagc acggacccgc agcccctcaa ggagcagccc 600
gccctcaatg actccagata ctgcctgagc agccgcctga gggtctcggc caccttctgg 660
cagaaccccc gcaaccactt ccgctgtcaa gtccagttct acgggctctc ggagaatgac 720
gagtggaccc aggatagggc caaacccgtc acccagatcg tcagcgccga ggcctggggt 780
agagcagact gtggctttac ctcggtgtcc taccagcaag gggtcctgtc tgccaccatc 840
ctctatgaga tcctgctagg gaaggccacc ctgtatgctg tgctggtcag cgcccttgtg 900
ttgatggcca tggtcaagag aaaggatttc 930
<210> 45
<211> 930
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 5 - (E6)29 - TCR бета, кодон-оптимизированный /
цистеин-модифицированный
<400> 45
atgggaatca ggctgctgtg ccgcgtcgca ttctgttttc tggccgtggg cctggtggac 60
gtgaaagtga ctcagagctc cagatacctg gtgaaaagga ccggcgagaa ggtctttctg 120
gaatgcgtgc aggacatgga tcacgagaat atgttctggt atcggcagga tccaggcctg 180
gggctgagac tgatctactt ttcctatgat gtgaagatga aagagaaggg cgacattccc 240
gaagggtact ccgtgtctcg cgagaagaaa gaacgattca gcctgatcct ggagagtgct 300
tcaaccaatc agacatccat gtatctgtgc gcatctagtc cttggggcga gacacaccag 360
ccacagcatt tcggagatgg cactcggctg agcatcctgg aagacctgaa caaagtgttc 420
ccccctgagg tcgccgtgtt cgaaccttca gaggcagaaa ttagccacac tcagaaggcc 480
accctggtgt gcctggccac tggcttcttt ccagaccacg tcgagctgtc ctggtgggtg 540
aatgggaaag aagtccatag tggagtgtgc accgacccac agcccctgaa ggagcagccc 600
gcactgaacg attccagata ctgcctgtca agccggctga gagtgtctgc cactttttgg 660
cagaaccctc gaaatcattt ccggtgtcag gtgcagtttt atggcctgag cgagaacgac 720
gaatggaccc aggatcgagc caaacctgtc acacagatcg tgtccgccga ggcttgggga 780
cgcgctgatt gcggcttcac aagcgtctcc taccagcagg gcgtgctgtc tgccaccatc 840
ctgtacgaaa ttctgctggg gaaggctaca ctgtatgccg tgctggtgag cgccctggtg 900
ctgatggcaa tggtgaaaag gaaggacttc 930
<210> 46
<211> 1853
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 5 - (E6)29 - TCR кодон-оптимизированный /
цистеин-модифицированный, полноразмерная последовательность
<400> 46
gcggccgcca ccatgggaat caggctgctg tgccgcgtcg cattctgttt tctggccgtg 60
ggcctggtgg acgtgaaagt gactcagagc tccagatacc tggtgaaaag gaccggcgag 120
aaggtctttc tggaatgcgt gcaggacatg gatcacgaga atatgttctg gtatcggcag 180
gatccaggcc tggggctgag actgatctac ttttcctatg atgtgaagat gaaagagaag 240
ggcgacattc ccgaagggta ctccgtgtct cgcgagaaga aagaacgatt cagcctgatc 300
ctggagagtg cttcaaccaa tcagacatcc atgtatctgt gcgcatctag tccttggggc 360
gagacacacc agccacagca tttcggagat ggcactcggc tgagcatcct ggaagacctg 420
aacaaagtgt tcccccctga ggtcgccgtg ttcgaacctt cagaggcaga aattagccac 480
actcagaagg ccaccctggt gtgcctggcc actggcttct ttccagacca cgtcgagctg 540
tcctggtggg tgaatgggaa agaagtccat agtggagtgt gcaccgaccc acagcccctg 600
aaggagcagc ccgcactgaa cgattccaga tactgcctgt caagccggct gagagtgtct 660
gccacttttt ggcagaaccc tcgaaatcat ttccggtgtc aggtgcagtt ttatggcctg 720
agcgagaacg acgaatggac ccaggatcga gccaaacctg tcacacagat cgtgtccgcc 780
gaggcttggg gacgcgctga ttgcggcttc acaagcgtct cctaccagca gggcgtgctg 840
tctgccacca tcctgtacga aattctgctg gggaaggcta cactgtatgc cgtgctggtg 900
agcgccctgg tgctgatggc aatggtgaaa aggaaggact tcgggtccgg agccacaaat 960
ttttctctgc tgaaacaggc tggcgatgtg gaggaaaacc ctgggccaat gagtctgtcc 1020
tctctgctga aggtggtcac tgcatcactg tggctgggac caggaatcgc acagaaaatt 1080
acccagacac agcctggcat gtttgtccag gagaaggaag ccgtgaccct ggactgtact 1140
tacgacacca gcgatcagtc ctacgggctg ttttggtata agcagccaag ttcaggagag 1200
atgatcttcc tgatctacca gggcagctat gacgagcaga acgctacaga aggcaggtat 1260
agcctgaatt tccagaaagc ccgcaagtcc gctaacctgg tcatctctgc cagtcagctg 1320
ggggattctg ccatgtactt ttgcgctatg agggagggaa ctggcaccag ctatggaaag 1380
ctgaccttcg ggcagggaac aatcctgact gtccatccca acattcagaa tccagaccct 1440
gccgtgtacc agctgcgaga cagtaaaagc tccgataaga gcgtgtgcct gttcacagac 1500
tttgattctc agactaacgt gagccagagc aaagacagtg atgtctatat taccgacaag 1560
tgcgtgctgg atatgcgcag catggacttt aaatccaact ctgcagtggc ctggtctaat 1620
aagagtgatt tcgcttgcgc aaacgccttt aacaattcaa tcattcccga ggataccttc 1680
tttccaagcc ccgaatctag ttgtgacgtg aaactggtgg agaagtcttt cgaaacagat 1740
actaacctga attttcagaa tctgagtgtc atcgggttcc ggattctgct gctgaaggtg 1800
gccggattca acctgctgat gaccctgaga ctgtggtcaa gctgaggcgc gcc 1853
<210> 47
<211> 610
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 5 - (E6)29 - TCR полноразмерная последовательность,
цистеин-модифицированный
<400> 47
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Ser Pro Trp Gly Glu Thr His Gln Pro Gln His Phe Gly Asp Gly Thr
115 120 125
Arg Leu Ser Ile Leu Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu
305 310 315 320
Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Ser Leu Ser
325 330 335
Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu Gly Pro Gly Ile
340 345 350
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
355 360 365
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln Ser Tyr
370 375 380
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
385 390 395 400
Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly Arg Tyr
405 410 415
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
420 425 430
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Arg Glu
435 440 445
Gly Thr Gly Thr Ser Tyr Gly Lys Leu Thr Phe Gly Gln Gly Thr Ile
450 455 460
Leu Thr Val His Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln
465 470 475 480
Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp
485 490 495
Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr
500 505 510
Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser
515 520 525
Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn
530 535 540
Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro
545 550 555 560
Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp
565 570 575
Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu
580 585 590
Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp
595 600 605
Ser Ser
610
<210> 48
<211> 253
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 6 - альфа нативный
<400> 48
Gly Glu Asp Val Glu Gln Ser Leu Phe Leu Ser Val Arg Glu Gly Asp
1 5 10 15
Ser Ser Val Ile Asn Cys Thr Tyr Thr Asp Ser Ser Ser Thr Tyr Leu
20 25 30
Tyr Trp Tyr Lys Gln Glu Pro Gly Ala Gly Leu Gln Leu Leu Thr Tyr
35 40 45
Ile Phe Ser Asn Met Asp Met Lys Gln Asp Gln Arg Leu Thr Val Leu
50 55 60
Leu Asn Lys Lys Asp Lys His Leu Ser Leu Arg Ile Ala Asp Thr Gln
65 70 75 80
Thr Gly Asp Ser Ala Ile Tyr Phe Cys Ala Glu Ser Ile Arg Gly Phe
85 90 95
Gly Asn Val Leu His Cys Gly Ser Gly Thr Gln Val Ile Val Leu Pro
100 105 110
His Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys
115 120 125
Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr
130 135 140
Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr
145 150 155 160
Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala
165 170 175
Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser
180 185 190
Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp
195 200 205
Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe
210 215 220
Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala
225 230 235 240
Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 49
<211> 253
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 6 - альфа, цистеин-модифицированный
<400> 49
Gly Glu Asp Val Glu Gln Ser Leu Phe Leu Ser Val Arg Glu Gly Asp
1 5 10 15
Ser Ser Val Ile Asn Cys Thr Tyr Thr Asp Ser Ser Ser Thr Tyr Leu
20 25 30
Tyr Trp Tyr Lys Gln Glu Pro Gly Ala Gly Leu Gln Leu Leu Thr Tyr
35 40 45
Ile Phe Ser Asn Met Asp Met Lys Gln Asp Gln Arg Leu Thr Val Leu
50 55 60
Leu Asn Lys Lys Asp Lys His Leu Ser Leu Arg Ile Ala Asp Thr Gln
65 70 75 80
Thr Gly Asp Ser Ala Ile Tyr Phe Cys Ala Glu Ser Ile Arg Gly Phe
85 90 95
Gly Asn Val Leu His Cys Gly Ser Gly Thr Gln Val Ile Val Leu Pro
100 105 110
His Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys
115 120 125
Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr
130 135 140
Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys
145 150 155 160
Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala
165 170 175
Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser
180 185 190
Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp
195 200 205
Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe
210 215 220
Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala
225 230 235 240
Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 50
<211> 813
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 6 - альфа нативный
<400> 50
atgaagacat ttgctggatt ttcgttcctg tttttgtggc tgcagctgga ctgtatgagt 60
agaggagagg atgtggagca gagtcttttc ctgagtgtcc gagagggaga cagctccgtt 120
ataaactgca cttacacaga cagctcctcc acctacttat actggtataa gcaagaacct 180
ggagcaggtc tccagttgct gacgtatatt ttttcaaata tggacatgaa acaagaccaa 240
agactcactg ttctattgaa taaaaaggat aaacatctgt ctctgcgcat tgcagacacc 300
cagactgggg actcagctat ctacttctgt gcagagagta taagaggctt tgggaatgtg 360
ctgcattgcg ggtccggcac tcaagtgatt gttttaccac atatccagaa ccctgaccct 420
gccgtgtacc agctgagaga ctctaaatcc agtgacaagt ctgtctgcct attcaccgat 480
tttgattctc aaacaaatgt gtcacaaagt aaggattctg atgtgtatat cacagacaaa 540
actgtgctag acatgaggtc tatggacttc aagagcaaca gtgctgtggc ctggagcaac 600
aaatctgact ttgcatgtgc aaacgccttc aacaacagca ttattccaga agacaccttc 660
ttccccagcc cagaaagttc ctgtgatgtc aagctggtcg agaaaagctt tgaaacagat 720
acgaacctaa actttcaaaa cctgtcagtg attgggttcc gaatcctcct cctgaaagtg 780
gccgggttta atctgctcat gacgctgcgg ctg 813
<210> 51
<211> 822
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 6 - альфа, кодон-оптимизированный/цистеин-модифицированный
<400> 51
atgaagacat ttgctggatt ttcgttcctg tttttgtggc tgcagctgga ctgtatgagt 60
agaggagagg atgtggagca gagtcttttc ctgagtgtcc gagagggaga cagctccgtt 120
ataaactgca cttacacaga cagctcctcc acctacttat actggtataa gcaagaacct 180
ggagcaggtc tccagttgct gacgtatatt ttttcaaata tggacatgaa acaagaccaa 240
agactcactg ttctattgaa taaaaaggat aaacatctgt ctctgcgcat tgcagacacc 300
cagactgggg actcagctat ctacttctgt gcagagagta taagaggctt tgggaatgtg 360
ctgcattgcg ggtccggcac tcaagtgatt gttttaccac atatccagaa ccctgaccct 420
gccgtgtacc agctgagaga ctctaaatcc agtgacaagt ctgtctgcct attcaccgat 480
tttgattctc aaacaaatgt gtcacaaagt aaggattctg atgtgtatat cacagacaaa 540
tgtgtgctag acatgaggtc tatggacttc aagagcaaca gtgctgtggc ctggagcaac 600
aaatctgact ttgcatgtgc aaacgccttc aacaacagca ttattccaga agacaccttc 660
ttccccagcc cagaaagttc ctgtgatgtc aagctggtcg agaaaagctt tgaaacagat 720
acgaacctaa actttcaaaa cctgtcagtg attgggttcc gaatcctcct cctgaaagtg 780
gccgggttta atctgctcat gacgctgcgg ctgtggtctt cc 822
<210> 52
<211> 292
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 6, TCR 12 - бета нативный
<400> 52
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Ser Thr Thr
85 90 95
Arg Ser Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu
115 120 125
Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys
130 135 140
Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val
145 150 155 160
Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro Leu
165 170 175
Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg
180 185 190
Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg
195 200 205
Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln
210 215 220
Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly
225 230 235 240
Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu
245 250 255
Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr
260 265 270
Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys
275 280 285
Asp Ser Arg Gly
290
<210> 53
<211> 292
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 6, TCR 12 - бета, цистеин-модифицированный
<400> 53
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Ser Thr Thr
85 90 95
Arg Ser Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu
115 120 125
Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys
130 135 140
Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val
145 150 155 160
Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro Leu
165 170 175
Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg
180 185 190
Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg
195 200 205
Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln
210 215 220
Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly
225 230 235 240
Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu
245 250 255
Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr
260 265 270
Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys
275 280 285
Asp Ser Arg Gly
290
<210> 54
<211> 933
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 6 - бета, кодон-оптимизированный/цистеин-модифицированный
<400> 54
atggatacct ggctcgtatg ctgggcaatt tttagtctct tgaaagcagg actcacagaa 60
cctgaagtca cccagactcc cagccatcag gtcacacaga tgggacagga agtgatcttg 120
cgctgtgtcc ccatctctaa tcacttatac ttctattggt acagacaaat cttggggcag 180
aaagtcgagt ttctggtttc cttttataat aatgaaatct cagagaagtc tgaaatattc 240
gatgatcaat tctcagttga aaggcctgat ggatcaaatt tcactctgaa gatccggtcc 300
acaaagctgg aggactcagc catgtacttc tgtgccagca caacgaggag ctcctacgag 360
cagtacttcg ggccgggcac caggctcacg gtcacagagg acctgaaaaa cgtgttccca 420
cccgaggtcg ctgtgtttga gccatcagaa gcagagatct cccacaccca aaaggccaca 480
ctggtatgcc tggccacagg cttctacccc gaccacgtgg agctgagctg gtgggtgaat 540
gggaaggagg tgcacagtgg ggtctgcaca gacccgcagc ccctcaagga gcagcccgcc 600
ctcaatgact ccagatactg cctgagcagc cgcctgaggg tctcggccac cttctggcag 660
aacccccgca accacttccg ctgtcaagtc cagttctacg ggctctcgga gaatgacgag 720
tggacccagg atagggccaa acctgtcacc cagatcgtca gcgccgaggc ctggggtaga 780
gcagactgtg gcttcacctc cgagtcttac cagcaagggg tcctgtctgc caccatcctc 840
tatgagatct tgctagggaa ggccaccttg tatgccgtgc tggtcagtgc cctcgtgctg 900
atggccatgg tcaagagaaa ggattccaga ggc 933
<210> 55
<211> 933
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 6 - бета нативный
<400> 55
atggatacct ggctcgtatg ctgggcaatt tttagtctct tgaaagcagg actcacagaa 60
cctgaagtca cccagactcc cagccatcag gtcacacaga tgggacagga agtgatcttg 120
cgctgtgtcc ccatctctaa tcacttatac ttctattggt acagacaaat cttggggcag 180
aaagtcgagt ttctggtttc cttttataat aatgaaatct cagagaagtc tgaaatattc 240
gatgatcaat tctcagttga aaggcctgat ggatcaaatt tcactctgaa gatccggtcc 300
acaaagctgg aggactcagc catgtacttc tgtgccagca caacgaggag ctcctacgag 360
cagtacttcg ggccgggcac caggctcacg gtcacagagg acctgaaaaa cgtgttccca 420
cccgaggtcg ctgtgtttga gccatcagaa gcagagatct cccacaccca aaaggccaca 480
ctggtatgcc tggccacagg cttctacccc gaccacgtgg agctgagctg gtgggtgaat 540
gggaaggagg tgcacagtgg ggtcagcaca gacccgcagc ccctcaagga gcagcccgcc 600
ctcaatgact ccagatactg cctgagcagc cgcctgaggg tctcggccac cttctggcag 660
aacccccgca accacttccg ctgtcaagtc cagttctacg ggctctcgga gaatgacgag 720
tggacccagg atagggccaa acctgtcacc cagatcgtca gcgccgaggc ctggggtaga 780
gcagactgtg gcttcacctc cgagtcttac cagcaagggg tcctgtctgc caccatcctc 840
tatgagatct tgctagggaa ggccaccttg tatgccgtgc tggtcagtgc cctcgtgctg 900
atggccatgg tcaagagaaa ggattccaga ggc 933
<210> 56
<211> 1844
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 6 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 56
gcggccgcca ccatggatac ctggctcgta tgctgggcaa tttttagtct cttgaaagca 60
ggactcacag aacctgaagt cacccagact cccagccatc aggtcacaca gatgggacag 120
gaagtgatct tgcgctgtgt ccccatctct aatcacttat acttctattg gtacagacaa 180
atcttggggc agaaagtcga gtttctggtt tccttttata ataatgaaat ctcagagaag 240
tctgaaatat tcgatgatca attctcagtt gaaaggcctg atggatcaaa tttcactctg 300
aagatccggt ccacaaagct ggaggactca gccatgtact tctgtgccag cacaacgagg 360
agctcctacg agcagtactt cgggccgggc accaggctca cggtcacaga ggacctgaaa 420
aacgtgttcc cacccgaggt cgctgtgttt gagccatcag aagcagagat ctcccacacc 480
caaaaggcca cactggtatg cctggccaca ggcttctacc ccgaccacgt ggagctgagc 540
tggtgggtga atgggaagga ggtgcacagt ggggtctgca cagacccgca gcccctcaag 600
gagcagcccg ccctcaatga ctccagatac tgcctgagca gccgcctgag ggtctcggcc 660
accttctggc agaacccccg caaccacttc cgctgtcaag tccagttcta cgggctctcg 720
gagaatgacg agtggaccca ggatagggcc aaacctgtca cccagatcgt cagcgccgag 780
gcctggggta gagcagactg tggcttcacc tccgagtctt accagcaagg ggtcctgtct 840
gccaccatcc tctatgagat cttgctaggg aaggccacct tgtatgccgt gctggtcagt 900
gccctcgtgc tgatggccat ggtcaagaga aaggattcca gaggcggatc cggagctacc 960
aacttctctc tgctgaaaca ggcaggcgat gtggaggaaa atcctgggcc aatgaagaca 1020
tttgctggat tttcgttcct gtttttgtgg ctgcagctgg actgtatgag tagaggagag 1080
gatgtggagc agagtctttt cctgagtgtc cgagagggag acagctccgt tataaactgc 1140
acttacacag acagctcctc cacctactta tactggtata agcaagaacc tggagcaggt 1200
ctccagttgc tgacgtatat tttttcaaat atggacatga aacaagacca aagactcact 1260
gttctattga ataaaaagga taaacatctg tctctgcgca ttgcagacac ccagactggg 1320
gactcagcta tctacttctg tgcagagagt ataagaggct ttgggaatgt gctgcattgc 1380
gggtccggca ctcaagtgat tgttttacca catatccaga accctgaccc tgccgtgtac 1440
cagctgagag actctaaatc cagtgacaag tctgtctgcc tattcaccga ttttgattct 1500
caaacaaatg tgtcacaaag taaggattct gatgtgtata tcacagacaa atgtgtgcta 1560
gacatgaggt ctatggactt caagagcaac agtgctgtgg cctggagcaa caaatctgac 1620
tttgcatgtg caaacgcctt caacaacagc attattccag aagacacctt cttccccagc 1680
ccagaaagtt cctgtgatgt caagctggtc gagaaaagct ttgaaacaga tacgaaccta 1740
aactttcaaa acctgtcagt gattgggttc cgaatcctcc tcctgaaagt ggccgggttt 1800
aatctgctca tgacgctgcg gctgtggtct tcctaaggcg cgcc 1844
<210> 57
<211> 607
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 6 Полноразмерная последовательность, цистеин-модифицированная
<400> 57
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ser Thr Thr Arg Ser Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser Leu
305 310 315 320
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Lys Thr
325 330 335
Phe Ala Gly Phe Ser Phe Leu Phe Leu Trp Leu Gln Leu Asp Cys Met
340 345 350
Ser Arg Gly Glu Asp Val Glu Gln Ser Leu Phe Leu Ser Val Arg Glu
355 360 365
Gly Asp Ser Ser Val Ile Asn Cys Thr Tyr Thr Asp Ser Ser Ser Thr
370 375 380
Tyr Leu Tyr Trp Tyr Lys Gln Glu Pro Gly Ala Gly Leu Gln Leu Leu
385 390 395 400
Thr Tyr Ile Phe Ser Asn Met Asp Met Lys Gln Asp Gln Arg Leu Thr
405 410 415
Val Leu Leu Asn Lys Lys Asp Lys His Leu Ser Leu Arg Ile Ala Asp
420 425 430
Thr Gln Thr Gly Asp Ser Ala Ile Tyr Phe Cys Ala Glu Ser Ile Arg
435 440 445
Gly Phe Gly Asn Val Leu His Cys Gly Ser Gly Thr Gln Val Ile Val
450 455 460
Leu Pro His Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp
465 470 475 480
Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser
485 490 495
Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp
500 505 510
Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala
515 520 525
Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn
530 535 540
Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser
545 550 555 560
Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu
565 570 575
Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys
580 585 590
Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600 605
<210> 58
<211> 254
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 7/ TCR 54 - (E7)11 - альфа нативный
<400> 58
Lys Asn Gln Val Glu Gln Ser Pro Gln Ser Leu Ile Ile Leu Glu Gly
1 5 10 15
Lys Asn Cys Thr Leu Gln Cys Asn Tyr Thr Val Ser Pro Phe Ser Asn
20 25 30
Leu Arg Trp Tyr Lys Gln Asp Thr Gly Arg Gly Pro Val Ser Leu Thr
35 40 45
Ile Met Thr Phe Ser Glu Asn Thr Lys Ser Asn Gly Arg Tyr Thr Ala
50 55 60
Thr Leu Asp Ala Asp Thr Lys Gln Ser Ser Leu His Ile Thr Ala Ser
65 70 75 80
Gln Leu Ser Asp Ser Ala Ser Tyr Ile Cys Val Val Ser Arg Asp Asn
85 90 95
Tyr Gly Gln Asn Phe Val Phe Gly Pro Gly Thr Arg Leu Ser Val Leu
100 105 110
Pro Tyr Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser
115 120 125
Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln
130 135 140
Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys
145 150 155 160
Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val
165 170 175
Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn
180 185 190
Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys
195 200 205
Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn
210 215 220
Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val
225 230 235 240
Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 59
<211> 254
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 7/ TCR 54 - (E7)11 - альфа, цистеин-модифицированный
<400> 59
Lys Asn Gln Val Glu Gln Ser Pro Gln Ser Leu Ile Ile Leu Glu Gly
1 5 10 15
Lys Asn Cys Thr Leu Gln Cys Asn Tyr Thr Val Ser Pro Phe Ser Asn
20 25 30
Leu Arg Trp Tyr Lys Gln Asp Thr Gly Arg Gly Pro Val Ser Leu Thr
35 40 45
Ile Met Thr Phe Ser Glu Asn Thr Lys Ser Asn Gly Arg Tyr Thr Ala
50 55 60
Thr Leu Asp Ala Asp Thr Lys Gln Ser Ser Leu His Ile Thr Ala Ser
65 70 75 80
Gln Leu Ser Asp Ser Ala Ser Tyr Ile Cys Val Val Ser Arg Asp Asn
85 90 95
Tyr Gly Gln Asn Phe Val Phe Gly Pro Gly Thr Arg Leu Ser Val Leu
100 105 110
Pro Tyr Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser
115 120 125
Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln
130 135 140
Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys
145 150 155 160
Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val
165 170 175
Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn
180 185 190
Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys
195 200 205
Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn
210 215 220
Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val
225 230 235 240
Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 60
<211> 816
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 7 - (E7)11 - альфа нативный
<400> 60
atgaaaaagc atctgacgac cttcttggtg attttgtggc tttattttta tagggggaat 60
ggcaaaaacc aagtggagca gagtcctcag tccctgatca tcctggaggg aaagaactgc 120
actcttcaat gcaattatac agtgagcccc ttcagcaact taaggtggta taagcaagat 180
actgggagag gtcctgtttc cctgacaatc atgactttca gtgagaacac aaagtcgaac 240
ggaagatata cagcaactct ggatgcagac acaaagcaaa gctctctgca catcacagcc 300
tcccagctca gcgattcagc ctcctacatc tgtgtggtga gccgggataa ctatggtcag 360
aattttgtct ttggtcccgg aaccagattg tccgtgctgc cctatatcca gaaccctgac 420
cctgccgtgt accagctgag agactctaaa tccagtgaca agtctgtctg cctattcacc 480
gattttgatt ctcaaacaaa tgtgtcacaa agtaaggatt ctgatgtgta tatcacagac 540
aaaactgtgc tagacatgag gtctatggac ttcaagagca acagtgctgt ggcctggagc 600
aacaaatctg actttgcatg tgcaaacgcc ttcaacaaca gcattattcc agaagacacc 660
ttcttcccca gcccagaaag ttcctgtgat gtcaagctgg tcgagaaaag ctttgaaaca 720
gatacgaacc taaactttca aaacctgtca gtgattgggt tccgaatcct cctcctgaaa 780
gtggccgggt ttaatctgct catgacgctg cggctg 816
<210> 61
<211> 825
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 7 - (E7)11 - альфа кодон-оптимизированный /
цистеин-модифицированный
<400> 61
atgaagaaac acctgaccac cttcctggtc atcctgtggc tgtacttcta cagagggaac 60
ggaaagaatc aggtggaaca gagtccacag tcactgatca ttctggaggg caaaaactgc 120
actctgcagt gtaattatac cgtgagccca ttttccaatc tgcgatggta caagcaggac 180
actggacgag gacccgtgag cctgaccatt atgacattct ccgagaacac caagtctaat 240
ggccgctata cagccactct ggacgctgat actaaacagt ctagtctgca tatcaccgcc 300
tctcagctgt ctgatagtgc ttcatatatt tgcgtggtca gtagggacaa ctacgggcag 360
aatttcgtgt ttggaccagg aacccgactg tccgtcctgc cttatatcca gaaccccgac 420
cctgccgtgt accagctgag ggactctaag tcaagcgata aaagcgtgtg cctgttcaca 480
gactttgatt cccagactaa tgtgagccag tccaaggact ctgacgtgta cattactgac 540
aaatgcgtcc tggatatgcg cagcatggac tttaagtcta acagtgcagt ggcctggtct 600
aacaagagtg atttcgcttg cgcaaacgcc tttaacaata gtatcattcc cgaagatact 660
ttctttccat cacccgagtc ctcttgtgac gtgaagctgg tcgaaaaatc attcgagacc 720
gatacaaacc tgaattttca gaacctgtct gtgatcgggt tccggattct gctgctgaag 780
gtcgccggat tcaatctgct gatgacactg agactgtgga gttca 825
<210> 62
<211> 290
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 7/ TCR 54 - (E7)11 - бета нативный
<400> 62
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Ile Thr Asp
85 90 95
Arg Thr Asn Tyr Gly Tyr Thr Phe Gly Ser Gly Thr Arg Leu Thr Val
100 105 110
Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val Phe Glu
115 120 125
Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys
130 135 140
Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp Trp Val
145 150 155 160
Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro Leu
165 170 175
Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg
180 185 190
Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg
195 200 205
Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln
210 215 220
Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly
225 230 235 240
Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly Val Leu
245 250 255
Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr
260 265 270
Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys
275 280 285
Asp Phe
290
<210> 63
<211> 290
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 7/ TCR 54 - (E7)11 - бета, цистеин-модифицированный
<400> 63
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Ile Thr Asp
85 90 95
Arg Thr Asn Tyr Gly Tyr Thr Phe Gly Ser Gly Thr Arg Leu Thr Val
100 105 110
Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val Phe Glu
115 120 125
Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys
130 135 140
Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp Trp Val
145 150 155 160
Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro Leu
165 170 175
Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg
180 185 190
Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg
195 200 205
Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln
210 215 220
Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly
225 230 235 240
Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly Val Leu
245 250 255
Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr
260 265 270
Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys
275 280 285
Asp Phe
290
<210> 64
<211> 927
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 7 - (E7)11 -бета нативный
<400> 64
atggatacct ggctcgtatg ctgggcaatt tttagtctct tgaaagcagg actcacagaa 60
cctgaagtca cccagactcc cagccatcag gtcacacaga tgggacagga agtgatcttg 120
cgctgtgtcc ccatctctaa tcacttatac ttctattggt acagacaaat cttggggcag 180
aaagtcgagt ttctggtttc cttttataat aatgaaatct cagagaagtc tgaaatattc 240
gatgatcaat tctcagttga aaggcctgat ggatcaaatt tcactctgaa gatccggtcc 300
acaaagctgg aggactcagc catgtacttc tgtgccatta cagaccgcac taactatggc 360
tacaccttcg gttcggggac caggttaacc gttgtagagg acctgaacaa ggtgttccca 420
cccgaggtcg ctgtgtttga gccatcagaa gcagagatct cccacaccca aaaggccaca 480
ctggtgtgcc tggccacagg cttcttccct gaccacgtgg agctgagctg gtgggtgaat 540
gggaaggagg tgcacagtgg ggtcagcacg gacccgcagc ccctcaagga gcagcccgcc 600
ctcaatgact ccagatactg cctgagcagc cgcctgaggg tctcggccac cttctggcag 660
aacccccgca accacttccg ctgtcaagtc cagttctacg ggctctcgga gaatgacgag 720
tggacccagg atagggccaa acccgtcacc cagatcgtca gcgccgaggc ctggggtaga 780
gcagactgtg gctttacctc ggtgtcctac cagcaagggg tcctgtctgc caccatcctc 840
tatgagatcc tgctagggaa ggccaccctg tatgctgtgc tggtcagcgc ccttgtgttg 900
atggccatgg tcaagagaaa ggatttc 927
<210> 65
<211> 927
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 7 - (E7)11 - бета кодон-оптимизированный /
цистеин-модифицированный
<400> 65
atggacacct ggctggtgtg ctgggcaatc tttagtctgc tgaaggccgg actgaccgag 60
cctgaagtga ctcagacccc atcccaccag gtcacacaga tgggccagga agtgatcctg 120
cggtgcgtgc caatttccaa ccatctgtac ttctattggt acagacagat tctgggccag 180
aaggtggagt tcctggtcag cttttataac aacgagatct cagaaaagag cgagattttc 240
gacgatcagt tttcagtgga aagacccgat gggagcaatt tcaccctgaa gatcaggagt 300
acaaaactgg aggattcagc aatgtacttt tgcgccatta ctgaccgcac caactatgga 360
tacaccttcg gctccgggac acgactgact gtggtcgagg acctgaataa ggtgttcccc 420
cctgaagtgg ctgtctttga gccttcagag gcagaaatca gccacacaca gaaagccacc 480
ctggtgtgcc tggctacagg cttctttcca gatcacgtgg aactgagctg gtgggtcaac 540
ggcaaggagg tgcattccgg ggtctgcact gacccacagc ccctgaaaga gcagcccgct 600
ctgaatgata gcaggtattg cctgagctcc cggctgagag tgtccgccac cttttggcag 660
aaccctagga atcatttccg ctgtcaggtg cagttttacg gcctgtctga aaacgacgag 720
tggacccagg atcgagctaa gcctgtgaca cagatcgtca gcgccgaagc ttgggggcgc 780
gcagactgcg gattcaccag cgtgtcctac cagcagggcg tcctgtccgc cacaatcctg 840
tatgagattc tgctggggaa ggctactctg tacgcagtgc tggtctctgc tctggtgctg 900
atggcaatgg tcaagcggaa agacttc 927
<210> 66
<211> 1841
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 7 - (E7)11 - кодон-оптимизированный /
цистеин-модифицированный, полноразмерная последовательность
<400> 66
gcggccgcca ccatggacac ctggctggtg tgctgggcaa tctttagtct gctgaaggcc 60
ggactgaccg agcctgaagt gactcagacc ccatcccacc aggtcacaca gatgggccag 120
gaagtgatcc tgcggtgcgt gccaatttcc aaccatctgt acttctattg gtacagacag 180
attctgggcc agaaggtgga gttcctggtc agcttttata acaacgagat ctcagaaaag 240
agcgagattt tcgacgatca gttttcagtg gaaagacccg atgggagcaa tttcaccctg 300
aagatcagga gtacaaaact ggaggattca gcaatgtact tttgcgccat tactgaccgc 360
accaactatg gatacacctt cggctccggg acacgactga ctgtggtcga ggacctgaat 420
aaggtgttcc cccctgaagt ggctgtcttt gagccttcag aggcagaaat cagccacaca 480
cagaaagcca ccctggtgtg cctggctaca ggcttctttc cagatcacgt ggaactgagc 540
tggtgggtca acggcaagga ggtgcattcc ggggtctgca ctgacccaca gcccctgaaa 600
gagcagcccg ctctgaatga tagcaggtat tgcctgagct cccggctgag agtgtccgcc 660
accttttggc agaaccctag gaatcatttc cgctgtcagg tgcagtttta cggcctgtct 720
gaaaacgacg agtggaccca ggatcgagct aagcctgtga cacagatcgt cagcgccgaa 780
gcttgggggc gcgcagactg cggattcacc agcgtgtcct accagcaggg cgtcctgtcc 840
gccacaatcc tgtatgagat tctgctgggg aaggctactc tgtacgcagt gctggtctct 900
gctctggtgc tgatggcaat ggtcaagcgg aaagacttcg gaagcggcgc aacaaacttt 960
tccctgctga aacaggccgg agatgtggag gaaaatcctg gcccaatgaa gaaacacctg 1020
accaccttcc tggtcatcct gtggctgtac ttctacagag ggaacggaaa gaatcaggtg 1080
gaacagagtc cacagtcact gatcattctg gagggcaaaa actgcactct gcagtgtaat 1140
tataccgtga gcccattttc caatctgcga tggtacaagc aggacactgg acgaggaccc 1200
gtgagcctga ccattatgac attctccgag aacaccaagt ctaatggccg ctatacagcc 1260
actctggacg ctgatactaa acagtctagt ctgcatatca ccgcctctca gctgtctgat 1320
agtgcttcat atatttgcgt ggtcagtagg gacaactacg ggcagaattt cgtgtttgga 1380
ccaggaaccc gactgtccgt cctgccttat atccagaacc ccgaccctgc cgtgtaccag 1440
ctgagggact ctaagtcaag cgataaaagc gtgtgcctgt tcacagactt tgattcccag 1500
actaatgtga gccagtccaa ggactctgac gtgtacatta ctgacaaatg cgtcctggat 1560
atgcgcagca tggactttaa gtctaacagt gcagtggcct ggtctaacaa gagtgatttc 1620
gcttgcgcaa acgcctttaa caatagtatc attcccgaag atactttctt tccatcaccc 1680
gagtcctctt gtgacgtgaa gctggtcgaa aaatcattcg agaccgatac aaacctgaat 1740
tttcagaacc tgtctgtgat cgggttccgg attctgctgc tgaaggtcgc cggattcaat 1800
ctgctgatga cactgagact gtggagttca tgaggcgcgc c 1841
<210> 67
<211> 606
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 7/ TCR 54 - (E7)11 - полноразмерная последовательность,
цистеин-модифицированный
<400> 67
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ile Thr Asp Arg Thr Asn Tyr Gly Tyr Thr Phe Gly Ser Gly Thr Arg
115 120 125
Leu Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Phe Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu Lys
305 310 315 320
Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Lys Lys His Leu
325 330 335
Thr Thr Phe Leu Val Ile Leu Trp Leu Tyr Phe Tyr Arg Gly Asn Gly
340 345 350
Lys Asn Gln Val Glu Gln Ser Pro Gln Ser Leu Ile Ile Leu Glu Gly
355 360 365
Lys Asn Cys Thr Leu Gln Cys Asn Tyr Thr Val Ser Pro Phe Ser Asn
370 375 380
Leu Arg Trp Tyr Lys Gln Asp Thr Gly Arg Gly Pro Val Ser Leu Thr
385 390 395 400
Ile Met Thr Phe Ser Glu Asn Thr Lys Ser Asn Gly Arg Tyr Thr Ala
405 410 415
Thr Leu Asp Ala Asp Thr Lys Gln Ser Ser Leu His Ile Thr Ala Ser
420 425 430
Gln Leu Ser Asp Ser Ala Ser Tyr Ile Cys Val Val Ser Arg Asp Asn
435 440 445
Tyr Gly Gln Asn Phe Val Phe Gly Pro Gly Thr Arg Leu Ser Val Leu
450 455 460
Pro Tyr Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser
465 470 475 480
Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln
485 490 495
Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys
500 505 510
Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val
515 520 525
Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn
530 535 540
Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys
545 550 555 560
Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn
565 570 575
Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val
580 585 590
Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600 605
<210> 68
<211> 253
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 8 - альфа нативный
<400> 68
Lys Gln Glu Val Thr Gln Ile Pro Ala Ala Leu Ser Val Pro Glu Gly
1 5 10 15
Glu Asn Leu Val Leu Asn Cys Ser Phe Thr Asp Ser Ala Ile Tyr Asn
20 25 30
Leu Gln Trp Phe Arg Gln Asp Pro Gly Lys Gly Leu Thr Ser Leu Leu
35 40 45
Leu Ile Gln Ser Ser Gln Arg Glu Gln Thr Ser Gly Arg Leu Asn Ala
50 55 60
Ser Leu Asp Lys Ser Ser Gly Arg Ser Thr Leu Tyr Ile Ala Ala Ser
65 70 75 80
Gln Pro Gly Asp Ser Ala Thr Tyr Leu Cys Ala Val Arg Pro Leu Gly
85 90 95
Asn Thr Pro Leu Val Phe Gly Lys Gly Thr Arg Leu Ser Val Ile Ala
100 105 110
Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys
115 120 125
Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr
130 135 140
Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr
145 150 155 160
Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala
165 170 175
Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser
180 185 190
Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp
195 200 205
Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe
210 215 220
Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala
225 230 235 240
Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 69
<211> 253
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 8 - альфа, цистеин-модифицированный
<400> 69
Lys Gln Glu Val Thr Gln Ile Pro Ala Ala Leu Ser Val Pro Glu Gly
1 5 10 15
Glu Asn Leu Val Leu Asn Cys Ser Phe Thr Asp Ser Ala Ile Tyr Asn
20 25 30
Leu Gln Trp Phe Arg Gln Asp Pro Gly Lys Gly Leu Thr Ser Leu Leu
35 40 45
Leu Ile Gln Ser Ser Gln Arg Glu Gln Thr Ser Gly Arg Leu Asn Ala
50 55 60
Ser Leu Asp Lys Ser Ser Gly Arg Ser Thr Leu Tyr Ile Ala Ala Ser
65 70 75 80
Gln Pro Gly Asp Ser Ala Thr Tyr Leu Cys Ala Val Arg Pro Leu Gly
85 90 95
Asn Thr Pro Leu Val Phe Gly Lys Gly Thr Arg Leu Ser Val Ile Ala
100 105 110
Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys
115 120 125
Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr
130 135 140
Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys
145 150 155 160
Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala
165 170 175
Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser
180 185 190
Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp
195 200 205
Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe
210 215 220
Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala
225 230 235 240
Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 70
<211> 807
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 8 - альфа нативный
<400> 70
atggagaccc tcttgggcct gcttatcctt tggctgcagc tgcaatgggt gagcagcaaa 60
caggaggtga cacagattcc tgcagctctg agtgtcccag aaggagaaaa cttggttctc 120
aactgcagtt tcactgatag cgctatttac aacctccagt ggtttaggca ggaccctggg 180
aaaggtctca catctctgtt gcttattcag tcaagtcaga gagagcaaac aagtggaaga 240
cttaatgcct cgctggataa atcatcagga cgtagtactt tatacattgc agcttctcag 300
cctggtgact cagccaccta cctctgtgct gtgaggcctc tcggaaacac acctcttgtc 360
tttggaaagg gcacaagact ttctgtgatt gcaaatatcc agaaccctga ccctgccgtg 420
taccagctga gagactctaa atccagtgac aagtctgtct gcctattcac cgattttgat 480
tctcaaacaa atgtgtcaca aagtaaggat tctgatgtgt atatcacaga caaaactgtg 540
ctagacatga ggtctatgga cttcaagagc aacagtgctg tggcctggag caacaaatct 600
gactttgcat gtgcaaacgc cttcaacaac agcattattc cagaagacac cttcttcccc 660
agcccagaaa gttcctgtga tgtcaagctg gtcgagaaaa gctttgaaac agatacgaac 720
ctaaactttc aaaacctgtc agtgattggg ttccgaatcc tcctcctgaa agtggccggg 780
tttaatctgc tcatgacgct gcggctc 807
<210> 71
<211> 816
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 8 - альфа, кодон-оптимизированный/цистеин-модифицированный
<400> 71
atggagaccc tcttgggcct gcttatcctt tggctgcagc tgcaatgggt gagcagcaaa 60
caggaggtga cacagattcc tgcagctctg agtgtcccag aaggagaaaa cttggttctc 120
aactgcagtt tcactgatag cgctatttac aacctccagt ggtttaggca ggaccctggg 180
aaaggtctca catctctgtt gcttattcag tcaagtcaga gagagcaaac aagtggaaga 240
cttaatgcct cgctggataa atcatcagga cgtagtactt tatacattgc agcttctcag 300
cctggtgact cagccaccta cctctgtgct gtgaggcctc tcggaaacac acctcttgtc 360
tttggaaagg gcacaagact ttctgtgatt gcaaatatcc agaaccctga ccctgccgtg 420
taccagctga gagactctaa atccagtgac aagtctgtct gcctattcac cgattttgat 480
tctcaaacaa atgtgtcaca aagtaaggat tctgatgtgt atatcacaga caaatgcgtg 540
ctagacatga ggtctatgga cttcaagagc aacagtgctg tggcctggag caacaaatct 600
gactttgcat gtgcaaacgc cttcaacaac agcattattc cagaagacac cttcttcccc 660
agcccagaaa gttcctgtga tgtcaagctg gtcgagaaaa gctttgaaac agatacgaac 720
ctaaactttc aaaacctgtc agtgattggg ttccgaatcc tcctcctgaa agtggccggg 780
tttaatctgc tcatgacgct gcggctctgg tcttcc 816
<210> 72
<211> 291
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 8 - бета нативный
<400> 72
Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys Arg Thr Gly Glu Lys
1 5 10 15
Val Phe Leu Glu Cys Val Gln Asp Met Asp His Glu Asn Met Phe Trp
20 25 30
Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu Ile Tyr Phe Ser Tyr
35 40 45
Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro Glu Gly Tyr Ser Val
50 55 60
Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile Leu Glu Ser Ala Ser
65 70 75 80
Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser Ser Leu Trp Gly Ala
85 90 95
Ser Thr Asp Thr Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val Leu
100 105 110
Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu Pro
115 120 125
Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys Leu
130 135 140
Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val Asn
145 150 155 160
Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro Leu Lys
165 170 175
Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg Leu
180 185 190
Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg Cys
195 200 205
Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln Asp
210 215 220
Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly Arg
225 230 235 240
Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu Ser
245 250 255
Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr Ala
260 265 270
Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys Asp
275 280 285
Ser Arg Gly
290
<210> 73
<211> 291
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 8 - бета, цистеин-модифицированный
<400> 73
Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys Arg Thr Gly Glu Lys
1 5 10 15
Val Phe Leu Glu Cys Val Gln Asp Met Asp His Glu Asn Met Phe Trp
20 25 30
Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu Ile Tyr Phe Ser Tyr
35 40 45
Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro Glu Gly Tyr Ser Val
50 55 60
Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile Leu Glu Ser Ala Ser
65 70 75 80
Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser Ser Leu Trp Gly Ala
85 90 95
Ser Thr Asp Thr Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val Leu
100 105 110
Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu Pro
115 120 125
Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys Leu
130 135 140
Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val Asn
145 150 155 160
Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro Leu Lys
165 170 175
Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg Leu
180 185 190
Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg Cys
195 200 205
Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln Asp
210 215 220
Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly Arg
225 230 235 240
Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu Ser
245 250 255
Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr Ala
260 265 270
Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys Asp
275 280 285
Ser Arg Gly
290
<210> 74
<211> 936
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 8 - бета нативный
<400> 74
atgggaatca ggctcctctg tcgtgtggcc ttttgtttcc tggctgtagg cctcgtagat 60
gtgaaagtaa cccagagctc gagatatcta gtcaaaagga cgggagagaa agtttttctg 120
gaatgtgtcc aggatatgga ccatgaaaat atgttctggt atcgacaaga cccaggtctg 180
gggctacggc tgatctattt ctcatatgat gttaaaatga aagaaaaagg agatattcct 240
gaggggtaca gtgtctctag agagaagaag gagcgcttct ccctgattct ggagtccgcc 300
agcaccaacc agacatctat gtacctctgt gccagcagtt tatggggggc tagcacagat 360
acgcagtatt ttggcccagg cacccggctg acagtgctcg aggacctgaa aaacgtgttc 420
ccacccgagg tcgctgtgtt tgagccatca gaagcagaga tctcccacac ccaaaaggcc 480
acactggtat gcctggccac aggcttctac cccgaccacg tggagctgag ctggtgggtg 540
aatgggaagg aggtgcacag tggggtcagc acagacccgc agcccctcaa ggagcagccc 600
gccctcaatg actccagata ctgcctgagc agccgcctga gggtctcggc caccttctgg 660
cagaaccccc gcaaccactt ccgctgtcaa gtccagttct acgggctctc ggagaatgac 720
gagtggaccc aggatagggc caaacctgtc acccagatcg tcagcgccga ggcctggggt 780
agagcagact gtggcttcac ctccgagtct taccagcaag gggtcctgtc tgccaccatc 840
ctctatgaga tcttgctagg gaaggccacc ttgtatgccg tgctggtcag tgccctcgtg 900
ctgatggcca tggtcaagag aaaggattcc agaggc 936
<210> 75
<211> 936
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 8 - бета, кодон-оптимизированный/цистеин-модифицированный
<400> 75
atgggaatca ggctcctctg tcgtgtggcc ttttgtttcc tggctgtagg cctcgtagat 60
gtgaaagtaa cccagagctc gagatatcta gtcaaaagga cgggagagaa agtttttctg 120
gaatgtgtcc aggatatgga ccatgaaaat atgttctggt atcgacaaga cccaggtctg 180
gggctacggc tgatctattt ctcatatgat gttaaaatga aagaaaaagg agatattcct 240
gaggggtaca gtgtctctag agagaagaag gagcgcttct ccctgattct ggagtccgcc 300
agcaccaacc agacatctat gtacctctgt gccagcagtt tatggggggc tagcacagat 360
acgcagtatt ttggcccagg cacccggctg acagtgctcg aggacctgaa aaacgtgttc 420
ccacccgagg tcgctgtgtt tgagccatca gaagcagaga tctcccacac ccaaaaggcc 480
acactggtat gcctggccac aggcttctac cccgaccacg tggagctgag ctggtgggtg 540
aatgggaagg aggtgcacag tggggtctgt acagacccgc agcccctcaa ggagcagccc 600
gccctcaatg actccagata ctgcctgagc agccgcctga gggtctcggc caccttctgg 660
cagaaccccc gcaaccactt ccgctgtcaa gtccagttct acgggctctc ggagaatgac 720
gagtggaccc aggatagggc caaacctgtc acccagatcg tcagcgccga ggcctggggt 780
agagcagact gtggcttcac ctccgagtct taccagcaag gggtcctgtc tgccaccatc 840
ctctatgaga tcttgctagg gaaggccacc ttgtatgccg tgctggtcag tgccctcgtg 900
ctgatggcca tggtcaagag aaaggattcc agaggc 936
<210> 76
<211> 1841
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 8 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 76
gcggccgcca ccatgggaat caggctcctc tgtcgtgtgg ccttttgttt cctggctgta 60
ggcctcgtag atgtgaaagt aacccagagc tcgagatatc tagtcaaaag gacgggagag 120
aaagtttttc tggaatgtgt ccaggatatg gaccatgaaa atatgttctg gtatcgacaa 180
gacccaggtc tggggctacg gctgatctat ttctcatatg atgttaaaat gaaagaaaaa 240
ggagatattc ctgaggggta cagtgtctct agagagaaga aggagcgctt ctccctgatt 300
ctggagtccg ccagcaccaa ccagacatct atgtacctct gtgccagcag tttatggggg 360
gctagcacag atacgcagta ttttggccca ggcacccggc tgacagtgct cgaggacctg 420
aaaaacgtgt tcccacccga ggtcgctgtg tttgagccat cagaagcaga gatctcccac 480
acccaaaagg ccacactggt atgcctggcc acaggcttct accccgacca cgtggagctg 540
agctggtggg tgaatgggaa ggaggtgcac agtggggtct gtacagaccc gcagcccctc 600
aaggagcagc ccgccctcaa tgactccaga tactgcctga gcagccgcct gagggtctcg 660
gccaccttct ggcagaaccc ccgcaaccac ttccgctgtc aagtccagtt ctacgggctc 720
tcggagaatg acgagtggac ccaggatagg gccaaacctg tcacccagat cgtcagcgcc 780
gaggcctggg gtagagcaga ctgtggcttc acctccgagt cttaccagca aggggtcctg 840
tctgccacca tcctctatga gatcttgcta gggaaggcca ccttgtatgc cgtgctggtc 900
agtgccctcg tgctgatggc catggtcaag agaaaggatt ccagaggcgg atccggagct 960
accaacttct ctctgctgaa acaggcaggc gatgtggagg aaaatcctgg gccaatggag 1020
accctcttgg gcctgcttat cctttggctg cagctgcaat gggtgagcag caaacaggag 1080
gtgacacaga ttcctgcagc tctgagtgtc ccagaaggag aaaacttggt tctcaactgc 1140
agtttcactg atagcgctat ttacaacctc cagtggttta ggcaggaccc tgggaaaggt 1200
ctcacatctc tgttgcttat tcagtcaagt cagagagagc aaacaagtgg aagacttaat 1260
gcctcgctgg ataaatcatc aggacgtagt actttataca ttgcagcttc tcagcctggt 1320
gactcagcca cctacctctg tgctgtgagg cctctcggaa acacacctct tgtctttgga 1380
aagggcacaa gactttctgt gattgcaaat atccagaacc ctgaccctgc cgtgtaccag 1440
ctgagagact ctaaatccag tgacaagtct gtctgcctat tcaccgattt tgattctcaa 1500
acaaatgtgt cacaaagtaa ggattctgat gtgtatatca cagacaaatg cgtgctagac 1560
atgaggtcta tggacttcaa gagcaacagt gctgtggcct ggagcaacaa atctgacttt 1620
gcatgtgcaa acgccttcaa caacagcatt attccagaag acaccttctt ccccagccca 1680
gaaagttcct gtgatgtcaa gctggtcgag aaaagctttg aaacagatac gaacctaaac 1740
tttcaaaacc tgtcagtgat tgggttccga atcctcctcc tgaaagtggc cgggtttaat 1800
ctgctcatga cgctgcggct ctggtcttcc taaggcgcgc c 1841
<210> 77
<211> 606
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 8 Полноразмерная последовательность, цистеин-модифицированная
<400> 77
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Ser Leu Trp Gly Ala Ser Thr Asp Thr Gln Tyr Phe Gly Pro Gly Thr
115 120 125
Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser
305 310 315 320
Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Glu
325 330 335
Thr Leu Leu Gly Leu Leu Ile Leu Trp Leu Gln Leu Gln Trp Val Ser
340 345 350
Ser Lys Gln Glu Val Thr Gln Ile Pro Ala Ala Leu Ser Val Pro Glu
355 360 365
Gly Glu Asn Leu Val Leu Asn Cys Ser Phe Thr Asp Ser Ala Ile Tyr
370 375 380
Asn Leu Gln Trp Phe Arg Gln Asp Pro Gly Lys Gly Leu Thr Ser Leu
385 390 395 400
Leu Leu Ile Gln Ser Ser Gln Arg Glu Gln Thr Ser Gly Arg Leu Asn
405 410 415
Ala Ser Leu Asp Lys Ser Ser Gly Arg Ser Thr Leu Tyr Ile Ala Ala
420 425 430
Ser Gln Pro Gly Asp Ser Ala Thr Tyr Leu Cys Ala Val Arg Pro Leu
435 440 445
Gly Asn Thr Pro Leu Val Phe Gly Lys Gly Thr Arg Leu Ser Val Ile
450 455 460
Ala Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser
465 470 475 480
Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln
485 490 495
Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys
500 505 510
Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val
515 520 525
Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn
530 535 540
Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys
545 550 555 560
Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn
565 570 575
Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val
580 585 590
Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600 605
<210> 78
<211> 258
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 9 - альфа нативный
<400> 78
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
1 5 10 15
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Pro Ser Tyr
20 25 30
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
35 40 45
Ile Tyr Gln Gly Ser Tyr Asp Gln Gln Asn Ala Thr Glu Gly Arg Tyr
50 55 60
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
65 70 75 80
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Arg Thr
85 90 95
Ala Gly Gly Thr Ser Tyr Gly Lys Leu Thr Phe Gly Gln Gly Thr Ile
100 105 110
Leu Thr Val His Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln
115 120 125
Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp
130 135 140
Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr
145 150 155 160
Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser
165 170 175
Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn
180 185 190
Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro
195 200 205
Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp
210 215 220
Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu
225 230 235 240
Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp
245 250 255
Ser Ser
<210> 79
<211> 258
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 9 - альфа, цистеин-модифицированный
<400> 79
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
1 5 10 15
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Pro Ser Tyr
20 25 30
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
35 40 45
Ile Tyr Gln Gly Ser Tyr Asp Gln Gln Asn Ala Thr Glu Gly Arg Tyr
50 55 60
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
65 70 75 80
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Arg Thr
85 90 95
Ala Gly Gly Thr Ser Tyr Gly Lys Leu Thr Phe Gly Gln Gly Thr Ile
100 105 110
Leu Thr Val His Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln
115 120 125
Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp
130 135 140
Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr
145 150 155 160
Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser
165 170 175
Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn
180 185 190
Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro
195 200 205
Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp
210 215 220
Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu
225 230 235 240
Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp
245 250 255
Ser Ser
<210> 80
<211> 825
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 9 - альфа нативный
<400> 80
atgtcacttt ctagcctgct gaaggtggtc acagcttcac tgtggctagg acctggcatt 60
gcccagaaga taactcaaac ccaaccagga atgttcgtgc aggaaaagga ggctgtgact 120
ctggactgca catatgacac cagtgatcca agttatggtc tattctggta caagcagccc 180
agcagtgggg aaatgatttt tcttatttat caggggtctt atgaccagca aaatgcaaca 240
gaaggtcgct actcattgaa tttccagaag gcaagaaaat ccgccaacct tgtcatctcc 300
gcttcacaac tgggggactc agcaatgtac ttctgtgcaa tgagaactgc tggtggtact 360
agctatggaa agctgacatt tggacaaggg accatcttga ctgtccatcc aaatatccag 420
aaccctgacc ctgccgtgta ccagctgaga gactctaaat ccagtgacaa gtctgtctgc 480
ctattcaccg attttgattc tcaaacaaat gtgtcacaaa gtaaggattc tgatgtgtat 540
atcacagaca aaactgtgct agacatgagg tctatggact tcaagagcaa cagtgctgtg 600
gcctggagca acaaatctga ctttgcatgt gcaaacgcct tcaacaacag cattattcca 660
gaagacacct tcttccccag cccagaaagt tcctgtgatg tcaagctggt cgagaaaagc 720
tttgaaacag atacgaacct aaactttcaa aacctgtcag tgattgggtt ccgaatcctc 780
ctcctgaaag tggccgggtt taatctgctc atgacgctgc ggctg 825
<210> 81
<211> 834
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 9 - альфа, кодон-оптимизированный/цистеин-модифицированный
<400> 81
atgtcacttt ctagcctgct gaaggtggtc acagcttcac tgtggctagg acctggcatt 60
gcccagaaga taactcaaac ccaaccagga atgttcgtgc aggaaaagga ggctgtgact 120
ctggactgca catatgacac cagtgatcca agttatggtc tattctggta caagcagccc 180
agcagtgggg aaatgatttt tcttatttat caggggtctt atgaccagca aaatgcaaca 240
gaaggtcgct actcattgaa tttccagaag gcaagaaaat ccgccaacct tgtcatctcc 300
gcttcacaac tgggggactc agcaatgtac ttctgtgcaa tgagaactgc tggtggtact 360
agctatggaa agctgacatt tggacaaggg accatcttga ctgtccatcc aaatatccag 420
aaccctgacc ctgccgtgta ccagctgaga gactctaaat ccagtgacaa gtctgtctgc 480
ctattcaccg attttgattc tcaaacaaat gtgtcacaaa gtaaggattc tgatgtgtat 540
atcacagaca aatgtgtgct agacatgagg tctatggact tcaagagcaa cagtgctgtg 600
gcctggagca acaaatctga ctttgcatgt gcaaacgcct tcaacaacag cattattcca 660
gaagacacct tcttccccag cccagaaagt tcctgtgatg tcaagctggt cgagaaaagc 720
tttgaaacag atacgaacct aaactttcaa aacctgtcag tgattgggtt ccgaatcctc 780
ctcctgaaag tggccgggtt taatctgctc atgacgctgc ggctgtggtc ttcc 834
<210> 82
<211> 292
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 9 - бета нативный
<400> 82
Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Arg Val Leu Lys Thr Gly
1 5 10 15
Gln Ser Met Thr Leu Leu Cys Ala Gln Asp Met Asn His Glu Tyr Met
20 25 30
Tyr Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu Ile His Tyr
35 40 45
Ser Val Gly Glu Gly Thr Thr Ala Lys Gly Glu Val Pro Asp Gly Tyr
50 55 60
Asn Val Ser Arg Leu Lys Lys Gln Asn Phe Leu Leu Gly Leu Glu Ser
65 70 75 80
Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser Ser Tyr Phe
85 90 95
Gly Thr Ala Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu
115 120 125
Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys
130 135 140
Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val
145 150 155 160
Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro Leu
165 170 175
Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg
180 185 190
Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg
195 200 205
Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln
210 215 220
Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly
225 230 235 240
Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu
245 250 255
Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr
260 265 270
Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys
275 280 285
Asp Ser Arg Gly
290
<210> 83
<211> 292
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 9 - бета, цистеин-модифицированный
<400> 83
Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Arg Val Leu Lys Thr Gly
1 5 10 15
Gln Ser Met Thr Leu Leu Cys Ala Gln Asp Met Asn His Glu Tyr Met
20 25 30
Tyr Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu Ile His Tyr
35 40 45
Ser Val Gly Glu Gly Thr Thr Ala Lys Gly Glu Val Pro Asp Gly Tyr
50 55 60
Asn Val Ser Arg Leu Lys Lys Gln Asn Phe Leu Leu Gly Leu Glu Ser
65 70 75 80
Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser Ser Tyr Phe
85 90 95
Gly Thr Ala Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu
115 120 125
Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys
130 135 140
Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val
145 150 155 160
Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro Leu
165 170 175
Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg
180 185 190
Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg
195 200 205
Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln
210 215 220
Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly
225 230 235 240
Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu
245 250 255
Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr
260 265 270
Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys
275 280 285
Asp Ser Arg Gly
290
<210> 84
<211> 933
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 9 - бета нативный
<400> 84
atgagcctcg ggctcctgtg ctgtggggcc ttttctctcc tgtgggcagg tccagtgaat 60
gctggtgtca ctcagacccc aaaattccgg gtcctgaaga caggacagag catgacactg 120
ctgtgtgccc aggatatgaa ccatgaatac atgtactggt atcgacaaga cccaggcatg 180
gggctgaggc tgattcatta ctcagttggt gagggtacaa ctgccaaagg agaggtccct 240
gatggctaca atgtctccag attaaaaaaa cagaatttcc tgctggggtt ggagtcggct 300
gctccctccc aaacatctgt gtacttctgt gccagcagtt acttcgggac agcctacgag 360
cagtacttcg ggccgggcac caggctcacg gtcacagagg acctgaaaaa cgtgttccca 420
cccgaggtcg ctgtgtttga gccatcagaa gcagagatct cccacaccca aaaggccaca 480
ctggtgtgcc tggccacagg cttctacccc gaccacgtgg agctgagctg gtgggtgaat 540
gggaaggagg tgcacagtgg ggtcagcaca gacccgcagc ccctcaagga gcagcccgcc 600
ctcaatgact ccagatactg cctgagcagc cgcctgaggg tctcggccac cttctggcag 660
aacccccgca accacttccg ctgtcaagtc cagttctacg ggctctcgga gaatgacgag 720
tggacccagg atagggccaa acctgtcacc cagatcgtca gcgccgaggc ctggggtaga 780
gcagactgtg gcttcacctc cgagtcttac cagcaagggg tcctgtctgc caccatcctc 840
tatgagatct tgctagggaa ggccaccttg tatgccgtgc tggtcagtgc cctcgtgctg 900
atggccatgg tcaagagaaa ggattccaga ggc 933
<210> 85
<211> 933
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 9 - бета
Кодон-оптимизированный/цистеин-модифицированный
<400> 85
atgagcctcg ggctcctgtg ctgtggggcc ttttctctcc tgtgggcagg tccagtgaat 60
gctggtgtca ctcagacccc aaaattccgg gtcctgaaga caggacagag catgacactg 120
ctgtgtgccc aggatatgaa ccatgaatac atgtactggt atcgacaaga cccaggcatg 180
gggctgaggc tgattcatta ctcagttggt gagggtacaa ctgccaaagg agaggtccct 240
gatggctaca atgtctccag attaaaaaaa cagaatttcc tgctggggtt ggagtcggct 300
gctccctccc aaacatctgt gtacttctgt gccagcagtt acttcgggac agcctacgag 360
cagtacttcg ggccgggcac caggctcacg gtcacagagg acctgaaaaa cgtgttccca 420
cccgaggtcg ctgtgtttga gccatcagaa gcagagatct cccacaccca aaaggccaca 480
ctggtgtgcc tggccacagg cttctacccc gaccacgtgg agctgagctg gtgggtgaat 540
gggaaggagg tgcacagtgg ggtctgtaca gacccgcagc ccctcaagga gcagcccgcc 600
ctcaatgact ccagatactg cctgagcagc cgcctgaggg tctcggccac cttctggcag 660
aacccccgca accacttccg ctgtcaagtc cagttctacg ggctctcgga gaatgacgag 720
tggacccagg atagggccaa acctgtcacc cagatcgtca gcgccgaggc ctggggtaga 780
gcagactgtg gcttcacctc cgagtcttac cagcaagggg tcctgtctgc caccatcctc 840
tatgagatct tgctagggaa ggccaccttg tatgccgtgc tggtcagtgc cctcgtgctg 900
atggccatgg tcaagagaaa ggattccaga ggc 933
<210> 86
<211> 1856
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 9 - Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 86
gcggccgcca ccatgagcct cgggctcctg tgctgtgggg ccttttctct cctgtgggca 60
ggtccagtga atgctggtgt cactcagacc ccaaaattcc gggtcctgaa gacaggacag 120
agcatgacac tgctgtgtgc ccaggatatg aaccatgaat acatgtactg gtatcgacaa 180
gacccaggca tggggctgag gctgattcat tactcagttg gtgagggtac aactgccaaa 240
ggagaggtcc ctgatggcta caatgtctcc agattaaaaa aacagaattt cctgctgggg 300
ttggagtcgg ctgctccctc ccaaacatct gtgtacttct gtgccagcag ttacttcggg 360
acagcctacg agcagtactt cgggccgggc accaggctca cggtcacaga ggacctgaaa 420
aacgtgttcc cacccgaggt cgctgtgttt gagccatcag aagcagagat ctcccacacc 480
caaaaggcca cactggtgtg cctggccaca ggcttctacc ccgaccacgt ggagctgagc 540
tggtgggtga atgggaagga ggtgcacagt ggggtctgta cagacccgca gcccctcaag 600
gagcagcccg ccctcaatga ctccagatac tgcctgagca gccgcctgag ggtctcggcc 660
accttctggc agaacccccg caaccacttc cgctgtcaag tccagttcta cgggctctcg 720
gagaatgacg agtggaccca ggatagggcc aaacctgtca cccagatcgt cagcgccgag 780
gcctggggta gagcagactg tggcttcacc tccgagtctt accagcaagg ggtcctgtct 840
gccaccatcc tctatgagat cttgctaggg aaggccacct tgtatgccgt gctggtcagt 900
gccctcgtgc tgatggccat ggtcaagaga aaggattcca gaggcggatc cggagctacc 960
aacttctctc tgctgaaaca ggcaggcgat gtggaggaaa atcctgggcc aatgtcactt 1020
tctagcctgc tgaaggtggt cacagcttca ctgtggctag gacctggcat tgcccagaag 1080
ataactcaaa cccaaccagg aatgttcgtg caggaaaagg aggctgtgac tctggactgc 1140
acatatgaca ccagtgatcc aagttatggt ctattctggt acaagcagcc cagcagtggg 1200
gaaatgattt ttcttattta tcaggggtct tatgaccagc aaaatgcaac agaaggtcgc 1260
tactcattga atttccagaa ggcaagaaaa tccgccaacc ttgtcatctc cgcttcacaa 1320
ctgggggact cagcaatgta cttctgtgca atgagaactg ctggtggtac tagctatgga 1380
aagctgacat ttggacaagg gaccatcttg actgtccatc caaatatcca gaaccctgac 1440
cctgccgtgt accagctgag agactctaaa tccagtgaca agtctgtctg cctattcacc 1500
gattttgatt ctcaaacaaa tgtgtcacaa agtaaggatt ctgatgtgta tatcacagac 1560
aaatgtgtgc tagacatgag gtctatggac ttcaagagca acagtgctgt ggcctggagc 1620
aacaaatctg actttgcatg tgcaaacgcc ttcaacaaca gcattattcc agaagacacc 1680
ttcttcccca gcccagaaag ttcctgtgat gtcaagctgg tcgagaaaag ctttgaaaca 1740
gatacgaacc taaactttca aaacctgtca gtgattgggt tccgaatcct cctcctgaaa 1800
gtggccgggt ttaatctgct catgacgctg cggctgtggt cttcctaagg cgcgcc 1856
<210> 87
<211> 611
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 9 - Полноразмерная последовательность, цистеин-модифицированная
<400> 87
Met Ser Leu Gly Leu Leu Cys Cys Gly Ala Phe Ser Leu Leu Trp Ala
1 5 10 15
Gly Pro Val Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Arg Val Leu
20 25 30
Lys Thr Gly Gln Ser Met Thr Leu Leu Cys Ala Gln Asp Met Asn His
35 40 45
Glu Tyr Met Tyr Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu
50 55 60
Ile His Tyr Ser Val Gly Glu Gly Thr Thr Ala Lys Gly Glu Val Pro
65 70 75 80
Asp Gly Tyr Asn Val Ser Arg Leu Lys Lys Gln Asn Phe Leu Leu Gly
85 90 95
Leu Glu Ser Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser
100 105 110
Ser Tyr Phe Gly Thr Ala Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser Leu
305 310 315 320
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Ser Leu
325 330 335
Ser Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu Gly Pro Gly
340 345 350
Ile Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu
355 360 365
Lys Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Pro Ser
370 375 380
Tyr Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe
385 390 395 400
Leu Ile Tyr Gln Gly Ser Tyr Asp Gln Gln Asn Ala Thr Glu Gly Arg
405 410 415
Tyr Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile
420 425 430
Ser Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Arg
435 440 445
Thr Ala Gly Gly Thr Ser Tyr Gly Lys Leu Thr Phe Gly Gln Gly Thr
450 455 460
Ile Leu Thr Val His Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr
465 470 475 480
Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr
485 490 495
Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val
500 505 510
Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys
515 520 525
Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala
530 535 540
Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser
545 550 555 560
Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr
565 570 575
Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile
580 585 590
Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu
595 600 605
Trp Ser Ser
610
<210> 88
<211> 258
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 10 - альфа нативный
<400> 88
Arg Lys Glu Val Glu Gln Asp Pro Gly Pro Phe Asn Val Pro Glu Gly
1 5 10 15
Ala Thr Val Ala Phe Asn Cys Thr Tyr Ser Asn Ser Ala Ser Gln Ser
20 25 30
Phe Phe Trp Tyr Arg Gln Asp Cys Arg Lys Glu Pro Lys Leu Leu Met
35 40 45
Ser Val Tyr Ser Ser Gly Asn Glu Asp Gly Arg Phe Thr Ala Gln Leu
50 55 60
Asn Arg Ala Ser Gln Tyr Ile Ser Leu Leu Ile Arg Asp Ser Lys Leu
65 70 75 80
Ser Asp Ser Ala Thr Tyr Leu Cys Val Val Asn Phe Pro Ser Arg Gly
85 90 95
Ala Gly Gly Thr Ser Tyr Gly Lys Leu Thr Phe Gly Gln Gly Thr Ile
100 105 110
Leu Thr Val His Pro Asn Ile Gln Lys Pro Asp Pro Ala Val Tyr Gln
115 120 125
Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp
130 135 140
Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr
145 150 155 160
Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser
165 170 175
Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn
180 185 190
Ala Phe Asn Asn Ser Ile Ile Pro Ala Asp Thr Phe Phe Pro Ser Pro
195 200 205
Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp
210 215 220
Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu
225 230 235 240
Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp
245 250 255
Ser Ser
<210> 89
<211> 258
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 10 - альфа, цистеин-модифицированный
<400> 89
Arg Lys Glu Val Glu Gln Asp Pro Gly Pro Phe Asn Val Pro Glu Gly
1 5 10 15
Ala Thr Val Ala Phe Asn Cys Thr Tyr Ser Asn Ser Ala Ser Gln Ser
20 25 30
Phe Phe Trp Tyr Arg Gln Asp Cys Arg Lys Glu Pro Lys Leu Leu Met
35 40 45
Ser Val Tyr Ser Ser Gly Asn Glu Asp Gly Arg Phe Thr Ala Gln Leu
50 55 60
Asn Arg Ala Ser Gln Tyr Ile Ser Leu Leu Ile Arg Asp Ser Lys Leu
65 70 75 80
Ser Asp Ser Ala Thr Tyr Leu Cys Val Val Asn Phe Pro Ser Arg Gly
85 90 95
Ala Gly Gly Thr Ser Tyr Gly Lys Leu Thr Phe Gly Gln Gly Thr Ile
100 105 110
Leu Thr Val His Pro Asn Ile Gln Lys Pro Asp Pro Ala Val Tyr Gln
115 120 125
Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp
130 135 140
Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr
145 150 155 160
Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser
165 170 175
Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn
180 185 190
Ala Phe Asn Asn Ser Ile Ile Pro Ala Asp Thr Phe Phe Pro Ser Pro
195 200 205
Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp
210 215 220
Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu
225 230 235 240
Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp
245 250 255
Ser Ser
<210> 90
<211> 831
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 10 - альфа нативный
<400> 90
atgatgatat ccttgagagt tttactggtg atcctgtggc ttcagttaag ctgggtttgg 60
agccaacgga aggaggtgga gcaggatcct ggacccttca atgttccaga gggagccact 120
gtcgctttca actgtactta cagcaacagt gcttctcagt ctttcttctg gtacagacag 180
gattgcagga aagaacctaa gttgctgatg tccgtatact ccagtggtaa tgaagatgga 240
aggtttacag cacagctcaa tagagccagc cagtatattt ccctgctcat cagagactcc 300
aagctcagtg attcagccac ctacctctgt gtggtgaact tcccttctcg gggtgctggt 360
ggtactagct atggaaagct gacatttgga caagggacca tcttgactgt ccatccaaat 420
atccagaagc ctgaccctgc cgtgtaccag ctgagagact ctaaatccag tgacaagtct 480
gtctgcctat tcaccgattt tgattctcaa acaaatgtgt cacaaagtaa ggattctgat 540
gtgtatatca cagacaaaac tgtgctagac atgaggtcta tggacttcaa gagcaacagt 600
gctgtggcct ggagcaacaa atctgacttt gcatgtgcaa acgccttcaa caacagcatt 660
attccagcag acaccttctt ccccagccca gaaagttcct gtgatgtcaa gctggtcgag 720
aaaagctttg aaacagatac gaacctaaac tttcaaaacc tgtcagtgat tgggttccga 780
atcctcctcc tgaaagtggc cgggtttaat ctgctcatga cgctgcggct g 831
<210> 91
<211> 840
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 10 - альфа, кодон-оптимизированный/цистеин-модифицированный
<400> 91
atgatgatat ccttgagagt tttactggtg atcctgtggc ttcagttaag ctgggtttgg 60
agccaacgga aggaggtgga gcaggatcct ggacccttca atgttccaga gggagccact 120
gtcgctttca actgtactta cagcaacagt gcttctcagt ctttcttctg gtacagacag 180
gattgcagga aagaacctaa gttgctgatg tccgtatact ccagtggtaa tgaagatgga 240
aggtttacag cacagctcaa tagagccagc cagtatattt ccctgctcat cagagactcc 300
aagctcagtg attcagccac ctacctctgt gtggtgaact tcccttctcg gggtgctggt 360
ggtactagct atggaaagct gacatttgga caagggacca tcttgactgt ccatccaaat 420
atccagaagc ctgaccctgc cgtgtaccag ctgagagact ctaaatccag tgacaagtct 480
gtctgcctat tcaccgattt tgattctcaa acaaatgtgt cacaaagtaa ggattctgat 540
gtgtatatca cagacaaatg tgtgctagac atgaggtcta tggacttcaa gagcaacagt 600
gctgtggcct ggagcaacaa atctgacttt gcatgtgcaa acgccttcaa caacagcatt 660
attccagcag acaccttctt ccccagccca gaaagttcct gtgatgtcaa gctggtcgag 720
aaaagctttg aaacagatac gaacctaaac tttcaaaacc tgtcagtgat tgggttccga 780
atcctcctcc tgaaagtggc cgggtttaat ctgctcatga cgctgcggct gtggtcttcc 840
<210> 92
<211> 291
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 10 - бета нативный
<400> 92
Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys Arg Thr Gly
1 5 10 15
Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His Glu Asn Met
20 25 30
Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu Ile Tyr Phe
35 40 45
Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro Glu Gly Tyr
50 55 60
Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile Leu Glu Ser
65 70 75 80
Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser Ser Leu Ser
85 90 95
Leu Thr Gly Asn Tyr Gly Tyr Thr Phe Gly Ser Gly Thr Arg Leu Thr
100 105 110
Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val Phe
115 120 125
Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val
130 135 140
Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp Trp
145 150 155 160
Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro
165 170 175
Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser
180 185 190
Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe
195 200 205
Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr
210 215 220
Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp
225 230 235 240
Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly Val
245 250 255
Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu
260 265 270
Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg
275 280 285
Lys Asp Phe
290
<210> 93
<211> 291
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 10 - бета, цистеин-модифицированный
<400> 93
Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys Arg Thr Gly
1 5 10 15
Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His Glu Asn Met
20 25 30
Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu Ile Tyr Phe
35 40 45
Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro Glu Gly Tyr
50 55 60
Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile Leu Glu Ser
65 70 75 80
Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser Ser Leu Ser
85 90 95
Leu Thr Gly Asn Tyr Gly Tyr Thr Phe Gly Ser Gly Thr Arg Leu Thr
100 105 110
Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val Phe
115 120 125
Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val
130 135 140
Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp Trp
145 150 155 160
Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro
165 170 175
Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser
180 185 190
Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe
195 200 205
Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr
210 215 220
Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp
225 230 235 240
Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly Val
245 250 255
Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu
260 265 270
Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg
275 280 285
Lys Asp Phe
290
<210> 94
<211> 930
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 10 - бета нативный
<400> 94
atgggaatca ggctcctctg tcgtgtggcc ttttgtttcc tggctgtagg cctcgtagat 60
gtgaaagtaa cccagagctc gagatatcta gtcaaaagga cgggagagaa agtttttctg 120
gaatgtgtcc aggatatgga ccatgaaaat atgttctggt atcgacaaga cccaggtctg 180
gggctacggc tgatctattt ctcatatgat gttaaaatga aagaaaaagg agatattcct 240
gaggggtaca gtgtctctag agagaagaag gagcgcttct ccctgattct ggagtccgcc 300
agcaccaacc agacatctat gtacctctgt gccagcagtt tatccctaac agggaactat 360
ggctacacct tcggttcggg gaccaggtta accgttgtag aggacctgaa caaggtgttc 420
ccacccgagg tcgctgtgtt tgagccatca gaagcagaga tctcccacac ccaaaaggcc 480
acactggtgt gcctggccac aggcttcttc cctgaccacg tggagctgag ctggtgggtg 540
aatgggaagg aggtgcacag tggggtcagc acggacccgc agcccctcaa ggagcagccc 600
gccctcaatg actccagata ctgcctgagc agccgcctga gggtctcggc caccttctgg 660
cagaaccccc gcaaccactt ccgctgtcaa gtccagttct acgggctctc ggagaatgac 720
gagtggaccc aggatagggc caaacccgtc acccagatcg tcagcgccga ggcctggggt 780
agagcagact gtggctttac ctcggtgtcc taccagcaag gggtcctgtc tgccaccatc 840
ctctatgaga tcctgctagg gaaggccacc ctgtatgctg tgctggtcag cgcccttgtg 900
ttgatggcca tggtcaagag aaaggatttc 930
<210> 95
<211> 930
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 10 - бета, кодон-оптимизированный/цистеин-модифицированный
<400> 95
atgggaatca ggctcctctg tcgtgtggcc ttttgtttcc tggctgtagg cctcgtagat 60
gtgaaagtaa cccagagctc gagatatcta gtcaaaagga cgggagagaa agtttttctg 120
gaatgtgtcc aggatatgga ccatgaaaat atgttctggt atcgacaaga cccaggtctg 180
gggctacggc tgatctattt ctcatatgat gttaaaatga aagaaaaagg agatattcct 240
gaggggtaca gtgtctctag agagaagaag gagcgcttct ccctgattct ggagtccgcc 300
agcaccaacc agacatctat gtacctctgt gccagcagtt tatccctaac agggaactat 360
ggctacacct tcggttcggg gaccaggtta accgttgtag aggacctgaa caaggtgttc 420
ccacccgagg tcgctgtgtt tgagccatca gaagcagaga tctcccacac ccaaaaggcc 480
acactggtgt gcctggccac aggcttcttc cctgaccacg tggagctgag ctggtgggtg 540
aatgggaagg aggtgcacag tggggtctgt acggacccgc agcccctcaa ggagcagccc 600
gccctcaatg actccagata ctgcctgagc agccgcctga gggtctcggc caccttctgg 660
cagaaccccc gcaaccactt ccgctgtcaa gtccagttct acgggctctc ggagaatgac 720
gagtggaccc aggatagggc caaacccgtc acccagatcg tcagcgccga ggcctggggt 780
agagcagact gtggctttac ctcggtgtcc taccagcaag gggtcctgtc tgccaccatc 840
ctctatgaga tcctgctagg gaaggccacc ctgtatgctg tgctggtcag cgcccttgtg 900
ttgatggcca tggtcaagag aaaggatttc 930
<210> 96
<211> 1859
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 10 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 96
gcggccgcca ccatgggaat caggctcctc tgtcgtgtgg ccttttgttt cctggctgta 60
ggcctcgtag atgtgaaagt aacccagagc tcgagatatc tagtcaaaag gacgggagag 120
aaagtttttc tggaatgtgt ccaggatatg gaccatgaaa atatgttctg gtatcgacaa 180
gacccaggtc tggggctacg gctgatctat ttctcatatg atgttaaaat gaaagaaaaa 240
ggagatattc ctgaggggta cagtgtctct agagagaaga aggagcgctt ctccctgatt 300
ctggagtccg ccagcaccaa ccagacatct atgtacctct gtgccagcag tttatcccta 360
acagggaact atggctacac cttcggttcg gggaccaggt taaccgttgt agaggacctg 420
aacaaggtgt tcccacccga ggtcgctgtg tttgagccat cagaagcaga gatctcccac 480
acccaaaagg ccacactggt gtgcctggcc acaggcttct tccctgacca cgtggagctg 540
agctggtggg tgaatgggaa ggaggtgcac agtggggtct gtacggaccc gcagcccctc 600
aaggagcagc ccgccctcaa tgactccaga tactgcctga gcagccgcct gagggtctcg 660
gccaccttct ggcagaaccc ccgcaaccac ttccgctgtc aagtccagtt ctacgggctc 720
tcggagaatg acgagtggac ccaggatagg gccaaacccg tcacccagat cgtcagcgcc 780
gaggcctggg gtagagcaga ctgtggcttt acctcggtgt cctaccagca aggggtcctg 840
tctgccacca tcctctatga gatcctgcta gggaaggcca ccctgtatgc tgtgctggtc 900
agcgcccttg tgttgatggc catggtcaag agaaaggatt tcggatccgg agctaccaac 960
ttctctctgc tgaaacaggc aggcgatgtg gaggaaaatc ctgggccaat gatgatatcc 1020
ttgagagttt tactggtgat cctgtggctt cagttaagct gggtttggag ccaacggaag 1080
gaggtggagc aggatcctgg acccttcaat gttccagagg gagccactgt cgctttcaac 1140
tgtacttaca gcaacagtgc ttctcagtct ttcttctggt acagacagga ttgcaggaaa 1200
gaacctaagt tgctgatgtc cgtatactcc agtggtaatg aagatggaag gtttacagca 1260
cagctcaata gagccagcca gtatatttcc ctgctcatca gagactccaa gctcagtgat 1320
tcagccacct acctctgtgt ggtgaacttc ccttctcggg gtgctggtgg tactagctat 1380
ggaaagctga catttggaca agggaccatc ttgactgtcc atccaaatat ccagaagcct 1440
gaccctgccg tgtaccagct gagagactct aaatccagtg acaagtctgt ctgcctattc 1500
accgattttg attctcaaac aaatgtgtca caaagtaagg attctgatgt gtatatcaca 1560
gacaaatgtg tgctagacat gaggtctatg gacttcaaga gcaacagtgc tgtggcctgg 1620
agcaacaaat ctgactttgc atgtgcaaac gccttcaaca acagcattat tccagcagac 1680
accttcttcc ccagcccaga aagttcctgt gatgtcaagc tggtcgagaa aagctttgaa 1740
acagatacga acctaaactt tcaaaacctg tcagtgattg ggttccgaat cctcctcctg 1800
aaagtggccg ggtttaatct gctcatgacg ctgcggctgt ggtcttccta aggcgcgcc 1859
<210> 97
<211> 612
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 10 Полноразмерная последовательность, цистеин-модифицированная
<400> 97
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Ser Leu Ser Leu Thr Gly Asn Tyr Gly Tyr Thr Phe Gly Ser Gly Thr
115 120 125
Arg Leu Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu
305 310 315 320
Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Met Ile Ser
325 330 335
Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu Ser Trp Val Trp
340 345 350
Ser Gln Arg Lys Glu Val Glu Gln Asp Pro Gly Pro Phe Asn Val Pro
355 360 365
Glu Gly Ala Thr Val Ala Phe Asn Cys Thr Tyr Ser Asn Ser Ala Ser
370 375 380
Gln Ser Phe Phe Trp Tyr Arg Gln Asp Cys Arg Lys Glu Pro Lys Leu
385 390 395 400
Leu Met Ser Val Tyr Ser Ser Gly Asn Glu Asp Gly Arg Phe Thr Ala
405 410 415
Gln Leu Asn Arg Ala Ser Gln Tyr Ile Ser Leu Leu Ile Arg Asp Ser
420 425 430
Lys Leu Ser Asp Ser Ala Thr Tyr Leu Cys Val Val Asn Phe Pro Ser
435 440 445
Arg Gly Ala Gly Gly Thr Ser Tyr Gly Lys Leu Thr Phe Gly Gln Gly
450 455 460
Thr Ile Leu Thr Val His Pro Asn Ile Gln Lys Pro Asp Pro Ala Val
465 470 475 480
Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe
485 490 495
Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp
500 505 510
Val Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe
515 520 525
Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys
530 535 540
Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Ala Asp Thr Phe Phe Pro
545 550 555 560
Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu
565 570 575
Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg
580 585 590
Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg
595 600 605
Leu Trp Ser Ser
610
<210> 98
<211> 253
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 11 - альфа нативный
<400> 98
Asp Ala Lys Thr Thr Gln Pro Asn Ser Met Glu Ser Asn Glu Glu Glu
1 5 10 15
Pro Val His Leu Pro Cys Asn His Ser Thr Ile Ser Gly Thr Asp Tyr
20 25 30
Ile His Trp Tyr Arg Gln Leu Pro Ser Gln Gly Pro Glu Tyr Val Ile
35 40 45
His Gly Leu Thr Ser Asn Val Asn Asn Arg Met Ala Ser Leu Ala Ile
50 55 60
Ala Glu Asp Arg Lys Ser Ser Thr Leu Ile Leu His Arg Ala Thr Leu
65 70 75 80
Arg Asp Ala Ala Val Tyr Tyr Cys Ile Leu Ser Ala His Ser Asn Ser
85 90 95
Gly Tyr Ala Leu Asn Phe Gly Lys Gly Thr Ser Leu Leu Val Thr Pro
100 105 110
His Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys
115 120 125
Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr
130 135 140
Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr
145 150 155 160
Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala
165 170 175
Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser
180 185 190
Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp
195 200 205
Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe
210 215 220
Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala
225 230 235 240
Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 99
<211> 253
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 11 - альфа, цистеин-модифицированный
<400> 99
Asp Ala Lys Thr Thr Gln Pro Asn Ser Met Glu Ser Asn Glu Glu Glu
1 5 10 15
Pro Val His Leu Pro Cys Asn His Ser Thr Ile Ser Gly Thr Asp Tyr
20 25 30
Ile His Trp Tyr Arg Gln Leu Pro Ser Gln Gly Pro Glu Tyr Val Ile
35 40 45
His Gly Leu Thr Ser Asn Val Asn Asn Arg Met Ala Ser Leu Ala Ile
50 55 60
Ala Glu Asp Arg Lys Ser Ser Thr Leu Ile Leu His Arg Ala Thr Leu
65 70 75 80
Arg Asp Ala Ala Val Tyr Tyr Cys Ile Leu Ser Ala His Ser Asn Ser
85 90 95
Gly Tyr Ala Leu Asn Phe Gly Lys Gly Thr Ser Leu Leu Val Thr Pro
100 105 110
His Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys
115 120 125
Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr
130 135 140
Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys
145 150 155 160
Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala
165 170 175
Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser
180 185 190
Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp
195 200 205
Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe
210 215 220
Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala
225 230 235 240
Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 100
<211> 801
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 11 - альфа нативный
<400> 100
atgaagttgg tgacaagcat tactgtactc ctatctttgg gtattatggg tgatgctaag 60
accacacagc caaattcaat ggagagtaac gaagaagagc ctgttcactt gccttgtaac 120
cactccacaa tcagtggaac tgattacata cattggtatc gacagcttcc ctcccagggt 180
ccagagtacg tgattcatgg tcttacaagc aatgtgaaca acagaatggc ctctctggca 240
atcgctgaag acagaaagtc cagtaccttg atcctgcacc gtgctacctt gagagatgct 300
gctgtgtact actgcatcct gagcgctcac tcaaattccg ggtatgcact caacttcggc 360
aaaggcacct cgctgttggt cacaccccat atccagaacc ctgaccctgc cgtgtaccag 420
ctgagagact ctaaatccag tgacaagtct gtctgcctat tcaccgattt tgattctcaa 480
acaaatgtgt cacaaagtaa ggattctgat gtgtatatca cagacaaaac tgtgctagac 540
atgaggtcta tggacttcaa gagcaacagt gctgtggcct ggagcaacaa atctgacttt 600
gcatgtgcaa acgccttcaa caacagcatt attccagaag acaccttctt ccccagccca 660
gaaagttcct gtgatgtcaa gctggtcgag aaaagctttg aaacagatac gaacctaaac 720
tttcaaaacc tgtcagtgat tgggttccga atcctcctcc tgaaagtggc cgggtttaat 780
ctgctcatga cgctgcggct g 801
<210> 101
<211> 810
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 11 - альфа, кодон-оптимизированный/цистеин-модифицированный
<400> 101
atgaagttgg tgacaagcat tactgtactc ctatctttgg gtattatggg tgatgctaag 60
accacacagc caaattcaat ggagagtaac gaagaagagc ctgttcactt gccttgtaac 120
cactccacaa tcagtggaac tgattacata cattggtatc gacagcttcc ctcccagggt 180
ccagagtacg tgattcatgg tcttacaagc aatgtgaaca acagaatggc ctctctggca 240
atcgctgaag acagaaagtc cagtaccttg atcctgcacc gtgctacctt gagagatgct 300
gctgtgtact actgcatcct gagcgctcac tcaaattccg ggtatgcact caacttcggc 360
aaaggcacct cgctgttggt cacaccccat atccagaacc ctgaccctgc cgtgtaccag 420
ctgagagact ctaaatccag tgacaagtct gtctgcctat tcaccgattt tgattctcaa 480
acaaatgtgt cacaaagtaa ggattctgat gtgtatatca cagacaaatg tgtgctagac 540
atgaggtcta tggacttcaa gagcaacagt gctgtggcct ggagcaacaa atctgacttt 600
gcatgtgcaa acgccttcaa caacagcatt attccagaag acaccttctt ccccagccca 660
gaaagttcct gtgatgtcaa gctggtcgag aaaagctttg aaacagatac gaacctaaac 720
tttcaaaacc tgtcagtgat tgggttccga atcctcctcc tgaaagtggc cgggtttaat 780
ctgctcatga cgctgcggct gtggtcttcc 810
<210> 102
<211> 297
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 11 - бета нативный
<400> 102
Ser Ala Val Ile Ser Gln Lys Pro Ser Arg Asp Ile Cys Gln Arg Gly
1 5 10 15
Thr Ser Leu Thr Ile Gln Cys Gln Val Asp Ser Gln Val Thr Met Met
20 25 30
Phe Trp Tyr Arg Gln Gln Pro Gly Gln Ser Leu Thr Leu Ile Ala Thr
35 40 45
Ala Asn Gln Gly Ser Glu Ala Thr Tyr Glu Ser Gly Phe Val Ile Asp
50 55 60
Lys Phe Pro Ile Ser Arg Pro Asn Leu Thr Phe Ser Thr Leu Thr Val
65 70 75 80
Ser Asn Met Ser Pro Glu Asp Ser Ser Ile Tyr Leu Cys Ser Val Val
85 90 95
Pro Trp Thr Arg Gly Gly Ser Thr Asp Thr Gln Tyr Phe Gly Pro Gly
100 105 110
Thr Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu
115 120 125
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
130 135 140
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu
145 150 155 160
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr
165 170 175
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
180 185 190
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
195 200 205
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
210 215 220
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
225 230 235 240
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr
245 250 255
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
260 265 270
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
275 280 285
Met Val Lys Arg Lys Asp Ser Arg Gly
290 295
<210> 103
<211> 297
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 11 - бета, цистеин-модифицированный
<400> 103
Ser Ala Val Ile Ser Gln Lys Pro Ser Arg Asp Ile Cys Gln Arg Gly
1 5 10 15
Thr Ser Leu Thr Ile Gln Cys Gln Val Asp Ser Gln Val Thr Met Met
20 25 30
Phe Trp Tyr Arg Gln Gln Pro Gly Gln Ser Leu Thr Leu Ile Ala Thr
35 40 45
Ala Asn Gln Gly Ser Glu Ala Thr Tyr Glu Ser Gly Phe Val Ile Asp
50 55 60
Lys Phe Pro Ile Ser Arg Pro Asn Leu Thr Phe Ser Thr Leu Thr Val
65 70 75 80
Ser Asn Met Ser Pro Glu Asp Ser Ser Ile Tyr Leu Cys Ser Val Val
85 90 95
Pro Trp Thr Arg Gly Gly Ser Thr Asp Thr Gln Tyr Phe Gly Pro Gly
100 105 110
Thr Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu
115 120 125
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
130 135 140
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu
145 150 155 160
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr
165 170 175
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
180 185 190
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
195 200 205
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
210 215 220
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
225 230 235 240
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr
245 250 255
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
260 265 270
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
275 280 285
Met Val Lys Arg Lys Asp Ser Arg Gly
290 295
<210> 104
<211> 936
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 11 - бета нативный
<400> 104
atgctgagtc ttctgctcct tctcctggga ctaggctctg tgttcagtgc tgtcatctct 60
caaaagccaa gcagggatat ctgtcaacgt ggaacctccc tgacgatcca gtgtcaagtc 120
gatagccaag tcaccatgat gttctggtac cgtcagcaac ctggacagag cctgacactg 180
atcgcaactg caaatcaggg ctctgaggcc acatatgaga gtggatttgt cattgacaag 240
tttcccatca gccgcccaaa cctaacattc tcaactctga ctgtgagcaa catgagccct 300
gaagacagca gcatatatct ctgcagcgtt gtcccttgga cgcgcggggg gagcacagat 360
acgcagtatt ttggcccagg cacccggctg acagtgctcg aggacctgaa aaacgtgttc 420
ccacccgagg tcgctgtgtt tgagccatca gaagcagaga tctcccacac ccaaaaggcc 480
acactggtgt gcctggccac aggcttctac cccgaccacg tggagctgag ctggtgggtg 540
aatgggaagg aggtgcacag tggggtcagc acagacccgc agcccctcaa ggagcagccc 600
gccctcaatg actccagata ctgcctgagc agccgcctga gggtctcggc caccttctgg 660
cagaaccccc gcaaccactt ccgctgtcaa gtccagttct acgggctctc ggagaatgac 720
gagtggaccc aggatagggc caaacctgtc acccagatcg tcagcgccga ggcctggggt 780
agagcagact gtggcttcac ctccgagtct taccagcaag gggtcctgtc tgccaccatc 840
ctctatgaga tcttgctagg gaaggccacc ttgtatgccg tgctggtcag tgccctcgtg 900
ctgatggcca tggtcaagag aaaggattcc agaggc 936
<210> 105
<211> 936
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 11 - бета, кодон-оптимизированный/цистеин-модифицированный
<400> 105
atgctgagtc ttctgctcct tctcctggga ctaggctctg tgttcagtgc tgtcatctct 60
caaaagccaa gcagggatat ctgtcaacgt ggaacctccc tgacgatcca gtgtcaagtc 120
gatagccaag tcaccatgat gttctggtac cgtcagcaac ctggacagag cctgacactg 180
atcgcaactg caaatcaggg ctctgaggcc acatatgaga gtggatttgt cattgacaag 240
tttcccatca gccgcccaaa cctaacattc tcaactctga ctgtgagcaa catgagccct 300
gaagacagca gcatatatct ctgcagcgtt gtcccttgga cgcgcggggg gagcacagat 360
acgcagtatt ttggcccagg cacccggctg acagtgctcg aggacctgaa aaacgtgttc 420
ccacccgagg tcgctgtgtt tgagccatca gaagcagaga tctcccacac ccaaaaggcc 480
acactggtgt gcctggccac aggcttctac cccgaccacg tggagctgag ctggtgggtg 540
aatgggaagg aggtgcacag tggggtctgt acagacccgc agcccctcaa ggagcagccc 600
gccctcaatg actccagata ctgcctgagc agccgcctga gggtctcggc caccttctgg 660
cagaaccccc gcaaccactt ccgctgtcaa gtccagttct acgggctctc ggagaatgac 720
gagtggaccc aggatagggc caaacctgtc acccagatcg tcagcgccga ggcctggggt 780
agagcagact gtggcttcac ctccgagtct taccagcaag gggtcctgtc tgccaccatc 840
ctctatgaga tcttgctagg gaaggccacc ttgtatgccg tgctggtcag tgccctcgtg 900
ctgatggcca tggtcaagag aaaggattcc agaggc 936
<210> 106
<211> 1835
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 11 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 106
gcggccgcca ccatgctgag tcttctgctc cttctcctgg gactaggctc tgtgttcagt 60
gctgtcatct ctcaaaagcc aagcagggat atctgtcaac gtggaacctc cctgacgatc 120
cagtgtcaag tcgatagcca agtcaccatg atgttctggt accgtcagca acctggacag 180
agcctgacac tgatcgcaac tgcaaatcag ggctctgagg ccacatatga gagtggattt 240
gtcattgaca agtttcccat cagccgccca aacctaacat tctcaactct gactgtgagc 300
aacatgagcc ctgaagacag cagcatatat ctctgcagcg ttgtcccttg gacgcgcggg 360
gggagcacag atacgcagta ttttggccca ggcacccggc tgacagtgct cgaggacctg 420
aaaaacgtgt tcccacccga ggtcgctgtg tttgagccat cagaagcaga gatctcccac 480
acccaaaagg ccacactggt gtgcctggcc acaggcttct accccgacca cgtggagctg 540
agctggtggg tgaatgggaa ggaggtgcac agtggggtct gtacagaccc gcagcccctc 600
aaggagcagc ccgccctcaa tgactccaga tactgcctga gcagccgcct gagggtctcg 660
gccaccttct ggcagaaccc ccgcaaccac ttccgctgtc aagtccagtt ctacgggctc 720
tcggagaatg acgagtggac ccaggatagg gccaaacctg tcacccagat cgtcagcgcc 780
gaggcctggg gtagagcaga ctgtggcttc acctccgagt cttaccagca aggggtcctg 840
tctgccacca tcctctatga gatcttgcta gggaaggcca ccttgtatgc cgtgctggtc 900
agtgccctcg tgctgatggc catggtcaag agaaaggatt ccagaggcgg atccggagct 960
accaacttct ctctgctgaa acaggcaggc gatgtggagg aaaatcctgg gccaatgaag 1020
ttggtgacaa gcattactgt actcctatct ttgggtatta tgggtgatgc taagaccaca 1080
cagccaaatt caatggagag taacgaagaa gagcctgttc acttgccttg taaccactcc 1140
acaatcagtg gaactgatta catacattgg tatcgacagc ttccctccca gggtccagag 1200
tacgtgattc atggtcttac aagcaatgtg aacaacagaa tggcctctct ggcaatcgct 1260
gaagacagaa agtccagtac cttgatcctg caccgtgcta ccttgagaga tgctgctgtg 1320
tactactgca tcctgagcgc tcactcaaat tccgggtatg cactcaactt cggcaaaggc 1380
acctcgctgt tggtcacacc ccatatccag aaccctgacc ctgccgtgta ccagctgaga 1440
gactctaaat ccagtgacaa gtctgtctgc ctattcaccg attttgattc tcaaacaaat 1500
gtgtcacaaa gtaaggattc tgatgtgtat atcacagaca aatgtgtgct agacatgagg 1560
tctatggact tcaagagcaa cagtgctgtg gcctggagca acaaatctga ctttgcatgt 1620
gcaaacgcct tcaacaacag cattattcca gaagacacct tcttccccag cccagaaagt 1680
tcctgtgatg tcaagctggt cgagaaaagc tttgaaacag atacgaacct aaactttcaa 1740
aacctgtcag tgattgggtt ccgaatcctc ctcctgaaag tggccgggtt taatctgctc 1800
atgacgctgc ggctgtggtc ttcctaaggc gcgcc 1835
<210> 107
<211> 604
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 11 Полноразмерная последовательность, цистеин-модифицированная
<400> 107
Met Leu Ser Leu Leu Leu Leu Leu Leu Gly Leu Gly Ser Val Phe Ser
1 5 10 15
Ala Val Ile Ser Gln Lys Pro Ser Arg Asp Ile Cys Gln Arg Gly Thr
20 25 30
Ser Leu Thr Ile Gln Cys Gln Val Asp Ser Gln Val Thr Met Met Phe
35 40 45
Trp Tyr Arg Gln Gln Pro Gly Gln Ser Leu Thr Leu Ile Ala Thr Ala
50 55 60
Asn Gln Gly Ser Glu Ala Thr Tyr Glu Ser Gly Phe Val Ile Asp Lys
65 70 75 80
Phe Pro Ile Ser Arg Pro Asn Leu Thr Phe Ser Thr Leu Thr Val Ser
85 90 95
Asn Met Ser Pro Glu Asp Ser Ser Ile Tyr Leu Cys Ser Val Val Pro
100 105 110
Trp Thr Arg Gly Gly Ser Thr Asp Thr Gln Tyr Phe Gly Pro Gly Thr
115 120 125
Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser
305 310 315 320
Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Lys
325 330 335
Leu Val Thr Ser Ile Thr Val Leu Leu Ser Leu Gly Ile Met Gly Asp
340 345 350
Ala Lys Thr Thr Gln Pro Asn Ser Met Glu Ser Asn Glu Glu Glu Pro
355 360 365
Val His Leu Pro Cys Asn His Ser Thr Ile Ser Gly Thr Asp Tyr Ile
370 375 380
His Trp Tyr Arg Gln Leu Pro Ser Gln Gly Pro Glu Tyr Val Ile His
385 390 395 400
Gly Leu Thr Ser Asn Val Asn Asn Arg Met Ala Ser Leu Ala Ile Ala
405 410 415
Glu Asp Arg Lys Ser Ser Thr Leu Ile Leu His Arg Ala Thr Leu Arg
420 425 430
Asp Ala Ala Val Tyr Tyr Cys Ile Leu Ser Ala His Ser Asn Ser Gly
435 440 445
Tyr Ala Leu Asn Phe Gly Lys Gly Thr Ser Leu Leu Val Thr Pro His
450 455 460
Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser
465 470 475 480
Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn
485 490 495
Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys Val
500 505 510
Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp
515 520 525
Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile
530 535 540
Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val
545 550 555 560
Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln
565 570 575
Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly
580 585 590
Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600
<210> 108
<211> 936
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 12 - бета нативный
<400> 108
atggatacct ggctcgtatg ctgggcaatt tttagtctct tgaaagcagg actcacagaa 60
cctgaagtca cccagactcc cagccatcag gtcacacaga tgggacagga agtgatcttg 120
cgctgtgtcc ccatctctaa tcacttatac ttctattggt acagacaaat cttggggcag 180
aaagtcgagt ttctggtttc cttttataat aatgaaatct cagagaagtc tgaaatattc 240
gatgatcaat tctcagttga aaggcctgat ggatcaaatt tcactctgaa gatccggtcc 300
acaaagctgg aggactcagc catgtacttc tgtgccagca caacgaggag ctcctacgag 360
cagtacttcg ggccgggcac caggctcacg gtcacagagg acctgaaaaa cgtgttccca 420
cccgaggtcg ctgtgtttga gccatcagaa gcagagatct cccacaccca aaaggccaca 480
ctggtatgcc tggccacagg cttctacccc gaccacgtgg agctgagctg gtgggtgaat 540
gggaaggagg tgcacagtgg ggtcagcaca gacccgcagc ccctcaagga gcagcccgcc 600
ctcaatgact ccagatactg cctgagcagc cgcctgaggg tctcggccac cttctggcag 660
aacccccgca accacttccg ctgtcaagtc cagttctacg ggctctcgga gaatgacgag 720
tggacccagg atagggccaa acctgtcacc cagatcgtca gcgccgaggc ctggggtaga 780
gcagactgtg gcttcacctc cgagtcttac cagcaagggg tcctgtctgc caccatcctc 840
tatgagatct tgctagggaa ggccaccttg tatgccgtgc tggtcagtgc cctcgtgctg 900
atggccatgg tcaagagaaa ggattccaga ggctag 936
<210> 109
<211> 172
<212> Белок
<213> Mus musculus
<220>
<223> Мышиная константная последовательность бета
<400> 109
Asp Leu Arg Asn Val Thr Pro Pro Lys Val Ser Leu Phe Glu Pro Ser
1 5 10 15
Lys Ala Glu Ile Ala Asn Lys Gln Lys Ala Thr Leu Val Cys Leu Ala
20 25 30
Arg Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp Trp Val Asn Gly
35 40 45
Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Ala Tyr Lys Glu
50 55 60
Ser Asn Tyr Ser Tyr Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr
65 70 75 80
Phe Trp His Asn Pro Arg Asn His Phe Arg Cys Gln Val Gln Phe His
85 90 95
Gly Leu Ser Glu Glu Asp Lys Trp Pro Glu Gly Ser Pro Lys Pro Val
100 105 110
Thr Gln Asn Ile Ser Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Ile
115 120 125
Thr Ser Ala Ser Tyr His Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr
130 135 140
Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Gly
145 150 155 160
Leu Val Leu Met Ala Met Val Lys Arg Lys Asn Ser
165 170
<210> 110
<211> 312
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 8 - бета нативный
<400> 110
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Ser Leu Trp Gly Ala Ser Thr Asp Thr Gln Tyr Phe Gly Pro Gly Thr
115 120 125
Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 111
<211> 114
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 3 альфа, вариабельная область
<400> 111
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
1 5 10 15
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln Ser Tyr
20 25 30
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
35 40 45
Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly Arg Tyr
50 55 60
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
65 70 75 80
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Arg Glu
85 90 95
Gly Arg Gly Phe Lys Thr Ile Phe Gly Ala Gly Thr Arg Leu Phe Val
100 105 110
Lys Ala
<210> 112
<211> 115
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 3 бета, вариабельная область
<400> 112
Gly Ala Gly Val Ser Gln Ser Pro Arg Tyr Lys Val Ala Lys Arg Gly
1 5 10 15
Gln Asp Val Ala Leu Arg Cys Asp Pro Ile Ser Gly His Val Ser Leu
20 25 30
Phe Trp Tyr Gln Gln Ala Leu Gly Gln Gly Pro Glu Phe Leu Thr Tyr
35 40 45
Phe Gln Asn Glu Ala Gln Leu Asp Lys Ser Gly Leu Pro Ser Asp Arg
50 55 60
Phe Phe Ala Glu Arg Pro Glu Gly Ser Val Ser Thr Leu Lys Ile Gln
65 70 75 80
Arg Thr Gln Gln Glu Asp Ser Ala Val Tyr Leu Cys Ala Ser Ser His
85 90 95
Leu Ala Gly Phe Thr Gly Glu Leu Phe Phe Gly Glu Gly Ser Arg Leu
100 105 110
Thr Val Leu
115
<210> 113
<211> 113
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 4 - (E6)29 альфа, вариабельная область
<400> 113
Asp Ala Lys Thr Thr Gln Pro Asn Ser Met Glu Ser Asn Glu Glu Glu
1 5 10 15
Pro Val His Leu Pro Cys Asn His Ser Thr Ile Ser Gly Thr Asp Tyr
20 25 30
Ile His Trp Tyr Arg Gln Leu Pro Ser Gln Gly Pro Glu Tyr Val Ile
35 40 45
His Gly Leu Thr Ser Asn Val Asn Asn Arg Met Ala Ser Leu Ala Ile
50 55 60
Ala Glu Asp Arg Lys Ser Ser Thr Leu Ile Leu His Arg Ala Thr Leu
65 70 75 80
Arg Asp Ala Ala Val Tyr Tyr Cys Ile Leu Leu Val Ile Arg Gly Thr
85 90 95
Ser Tyr Gly Lys Leu Thr Phe Gly Gln Gly Thr Ile Leu Thr Val His
100 105 110
Pro
<210> 114
<211> 112
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 4 - (E6)29 бета, вариабельная область
<400> 114
Gly Val Ser Gln Asp Pro Arg His Lys Ile Thr Lys Arg Gly Gln Asn
1 5 10 15
Val Thr Phe Arg Cys Asp Pro Ile Ser Glu His Asn Arg Leu Tyr Trp
20 25 30
Tyr Arg Gln Thr Leu Gly Gln Gly Pro Glu Phe Leu Thr Tyr Phe Gln
35 40 45
Asn Glu Ala Gln Leu Glu Lys Ser Arg Leu Leu Ser Asp Arg Phe Ser
50 55 60
Ala Glu Arg Pro Lys Gly Ser Phe Ser Thr Leu Glu Ile Gln Arg Thr
65 70 75 80
Glu Gln Gly Asp Ser Ala Met Tyr Leu Cys Ala Ser Ser Pro Gly Gly
85 90 95
Gly Asn Thr Glu Ala Phe Phe Gly Gln Gly Thr Arg Leu Thr Val Val
100 105 110
<210> 115
<211> 117
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 5 - (E6)29 - TCR альфа, вариабельная область
<400> 115
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
1 5 10 15
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln Ser Tyr
20 25 30
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
35 40 45
Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly Arg Tyr
50 55 60
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
65 70 75 80
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Arg Glu
85 90 95
Gly Thr Gly Thr Ser Tyr Gly Lys Leu Thr Phe Gly Gln Gly Thr Ile
100 105 110
Leu Thr Val His Pro
115
<210> 116
<211> 112
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 5 - (E6)29 - TCR бета, вариабельная область
<400> 116
Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys Arg Thr Gly Glu Lys
1 5 10 15
Val Phe Leu Glu Cys Val Gln Asp Met Asp His Glu Asn Met Phe Trp
20 25 30
Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu Ile Tyr Phe Ser Tyr
35 40 45
Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro Glu Gly Tyr Ser Val
50 55 60
Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile Leu Glu Ser Ala Ser
65 70 75 80
Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser Ser Pro Trp Gly Glu
85 90 95
Thr His Gln Pro Gln His Phe Gly Asp Gly Thr Arg Leu Ser Ile Leu
100 105 110
<210> 117
<211> 112
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 6 альфа, вариабельная область
<400> 117
Gly Glu Asp Val Glu Gln Ser Leu Phe Leu Ser Val Arg Glu Gly Asp
1 5 10 15
Ser Ser Val Ile Asn Cys Thr Tyr Thr Asp Ser Ser Ser Thr Tyr Leu
20 25 30
Tyr Trp Tyr Lys Gln Glu Pro Gly Ala Gly Leu Gln Leu Leu Thr Tyr
35 40 45
Ile Phe Ser Asn Met Asp Met Lys Gln Asp Gln Arg Leu Thr Val Leu
50 55 60
Leu Asn Lys Lys Asp Lys His Leu Ser Leu Arg Ile Ala Asp Thr Gln
65 70 75 80
Thr Gly Asp Ser Ala Ile Tyr Phe Cys Ala Glu Ser Ile Arg Gly Phe
85 90 95
Gly Asn Val Leu His Cys Gly Ser Gly Thr Gln Val Ile Val Leu Pro
100 105 110
<210> 118
<211> 113
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 6, TCR 12 бета, вариабельная область
<400> 118
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Ser Thr Thr
85 90 95
Arg Ser Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Thr
<210> 119
<211> 113
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 7/ TCR 54 - (E7)11 - альфа, вариабельная область
<400> 119
Lys Asn Gln Val Glu Gln Ser Pro Gln Ser Leu Ile Ile Leu Glu Gly
1 5 10 15
Lys Asn Cys Thr Leu Gln Cys Asn Tyr Thr Val Ser Pro Phe Ser Asn
20 25 30
Leu Arg Trp Tyr Lys Gln Asp Thr Gly Arg Gly Pro Val Ser Leu Thr
35 40 45
Ile Met Thr Phe Ser Glu Asn Thr Lys Ser Asn Gly Arg Tyr Thr Ala
50 55 60
Thr Leu Asp Ala Asp Thr Lys Gln Ser Ser Leu His Ile Thr Ala Ser
65 70 75 80
Gln Leu Ser Asp Ser Ala Ser Tyr Ile Cys Val Val Ser Arg Asp Asn
85 90 95
Tyr Gly Gln Asn Phe Val Phe Gly Pro Gly Thr Arg Leu Ser Val Leu
100 105 110
Pro
<210> 120
<211> 113
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 7/ TCR 54 - (E7)11 - бета, вариабельная область
<400> 120
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Ile Thr Asp
85 90 95
Arg Thr Asn Tyr Gly Tyr Thr Phe Gly Ser Gly Thr Arg Leu Thr Val
100 105 110
Val
<210> 121
<211> 112
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 8 альфа, вариабельная область
<400> 121
Lys Gln Glu Val Thr Gln Ile Pro Ala Ala Leu Ser Val Pro Glu Gly
1 5 10 15
Glu Asn Leu Val Leu Asn Cys Ser Phe Thr Asp Ser Ala Ile Tyr Asn
20 25 30
Leu Gln Trp Phe Arg Gln Asp Pro Gly Lys Gly Leu Thr Ser Leu Leu
35 40 45
Leu Ile Gln Ser Ser Gln Arg Glu Gln Thr Ser Gly Arg Leu Asn Ala
50 55 60
Ser Leu Asp Lys Ser Ser Gly Arg Ser Thr Leu Tyr Ile Ala Ala Ser
65 70 75 80
Gln Pro Gly Asp Ser Ala Thr Tyr Leu Cys Ala Val Arg Pro Leu Gly
85 90 95
Asn Thr Pro Leu Val Phe Gly Lys Gly Thr Arg Leu Ser Val Ile Ala
100 105 110
<210> 122
<211> 112
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 8
бета, вариабельная область
<400> 122
Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys Arg Thr Gly Glu Lys
1 5 10 15
Val Phe Leu Glu Cys Val Gln Asp Met Asp His Glu Asn Met Phe Trp
20 25 30
Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu Ile Tyr Phe Ser Tyr
35 40 45
Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro Glu Gly Tyr Ser Val
50 55 60
Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile Leu Glu Ser Ala Ser
65 70 75 80
Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser Ser Leu Trp Gly Ala
85 90 95
Ser Thr Asp Thr Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val Leu
100 105 110
<210> 123
<211> 117
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 9 альфа, вариабельная область
<400> 123
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
1 5 10 15
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Pro Ser Tyr
20 25 30
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
35 40 45
Ile Tyr Gln Gly Ser Tyr Asp Gln Gln Asn Ala Thr Glu Gly Arg Tyr
50 55 60
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
65 70 75 80
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Arg Thr
85 90 95
Ala Gly Gly Thr Ser Tyr Gly Lys Leu Thr Phe Gly Gln Gly Thr Ile
100 105 110
Leu Thr Val His Pro
115
<210> 124
<211> 113
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 9 бета, вариабельная область
<400> 124
Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Arg Val Leu Lys Thr Gly
1 5 10 15
Gln Ser Met Thr Leu Leu Cys Ala Gln Asp Met Asn His Glu Tyr Met
20 25 30
Tyr Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu Ile His Tyr
35 40 45
Ser Val Gly Glu Gly Thr Thr Ala Lys Gly Glu Val Pro Asp Gly Tyr
50 55 60
Asn Val Ser Arg Leu Lys Lys Gln Asn Phe Leu Leu Gly Leu Glu Ser
65 70 75 80
Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser Ser Tyr Phe
85 90 95
Gly Thr Ala Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Thr
<210> 125
<211> 117
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 10 альфа, вариабельная область
<400> 125
Arg Lys Glu Val Glu Gln Asp Pro Gly Pro Phe Asn Val Pro Glu Gly
1 5 10 15
Ala Thr Val Ala Phe Asn Cys Thr Tyr Ser Asn Ser Ala Ser Gln Ser
20 25 30
Phe Phe Trp Tyr Arg Gln Asp Cys Arg Lys Glu Pro Lys Leu Leu Met
35 40 45
Ser Val Tyr Ser Ser Gly Asn Glu Asp Gly Arg Phe Thr Ala Gln Leu
50 55 60
Asn Arg Ala Ser Gln Tyr Ile Ser Leu Leu Ile Arg Asp Ser Lys Leu
65 70 75 80
Ser Asp Ser Ala Thr Tyr Leu Cys Val Val Asn Phe Pro Ser Arg Gly
85 90 95
Ala Gly Gly Thr Ser Tyr Gly Lys Leu Thr Phe Gly Gln Gly Thr Ile
100 105 110
Leu Thr Val His Pro
115
<210> 126
<211> 112
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 10 бета, вариабельная область
<400> 126
Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys Arg Thr Gly Glu Lys
1 5 10 15
Val Phe Leu Glu Cys Val Gln Asp Met Asp His Glu Asn Met Phe Trp
20 25 30
Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu Ile Tyr Phe Ser Tyr
35 40 45
Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro Glu Gly Tyr Ser Val
50 55 60
Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile Leu Glu Ser Ala Ser
65 70 75 80
Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser Ser Leu Ser Leu Thr
85 90 95
Gly Asn Tyr Gly Tyr Thr Phe Gly Ser Gly Thr Arg Leu Thr Val Val
100 105 110
<210> 127
<211> 112
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 11 альфа, вариабельная область
<400> 127
Asp Ala Lys Thr Thr Gln Pro Asn Ser Met Glu Ser Asn Glu Glu Glu
1 5 10 15
Pro Val His Leu Pro Cys Asn His Ser Thr Ile Ser Gly Thr Asp Tyr
20 25 30
Ile His Trp Tyr Arg Gln Leu Pro Ser Gln Gly Pro Glu Tyr Val Ile
35 40 45
His Gly Leu Thr Ser Asn Val Asn Asn Arg Met Ala Ser Leu Ala Ile
50 55 60
Ala Glu Asp Arg Lys Ser Ser Thr Leu Ile Leu His Arg Ala Thr Leu
65 70 75 80
Arg Asp Ala Ala Val Tyr Tyr Cys Ile Leu Ser Ala His Ser Asn Ser
85 90 95
Gly Tyr Ala Leu Asn Phe Gly Lys Gly Thr Ser Leu Leu Val Thr Pro
100 105 110
<210> 128
<211> 118
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 11 бета, вариабельная область
<400> 128
Ser Ala Val Ile Ser Gln Lys Pro Ser Arg Asp Ile Cys Gln Arg Gly
1 5 10 15
Thr Ser Leu Thr Ile Gln Cys Gln Val Asp Ser Gln Val Thr Met Met
20 25 30
Phe Trp Tyr Arg Gln Gln Pro Gly Gln Ser Leu Thr Leu Ile Ala Thr
35 40 45
Ala Asn Gln Gly Ser Glu Ala Thr Tyr Glu Ser Gly Phe Val Ile Asp
50 55 60
Lys Phe Pro Ile Ser Arg Pro Asn Leu Thr Phe Ser Thr Leu Thr Val
65 70 75 80
Ser Asn Met Ser Pro Glu Asp Ser Ser Ile Tyr Leu Cys Ser Val Val
85 90 95
Pro Trp Thr Arg Gly Gly Ser Thr Asp Thr Gln Tyr Phe Gly Pro Gly
100 105 110
Thr Arg Leu Thr Val Leu
115
<210> 129
<211> 312
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 8 - бета, цистеин-модифицированный
<400> 129
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Ser Leu Trp Gly Ala Ser Thr Asp Thr Gln Tyr Phe Gly Pro Gly Thr
115 120 125
Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 130
<211> 278
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 9 - альфа нативный
<400> 130
Met Ser Leu Ser Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu
1 5 10 15
Gly Pro Gly Ile Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe
20 25 30
Val Gln Glu Lys Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser
35 40 45
Asp Pro Ser Tyr Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu
50 55 60
Met Ile Phe Leu Ile Tyr Gln Gly Ser Tyr Asp Gln Gln Asn Ala Thr
65 70 75 80
Glu Gly Arg Tyr Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn
85 90 95
Leu Val Ile Ser Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys
100 105 110
Ala Met Arg Thr Ala Gly Gly Thr Ser Tyr Gly Lys Leu Thr Phe Gly
115 120 125
Gln Gly Thr Ile Leu Thr Val His Pro Asn Ile Gln Asn Pro Asp Pro
130 135 140
Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys
145 150 155 160
Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp
165 170 175
Ser Asp Val Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met
180 185 190
Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe
195 200 205
Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe
210 215 220
Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser
225 230 235 240
Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly
245 250 255
Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr
260 265 270
Leu Arg Leu Trp Ser Ser
275
<210> 131
<211> 278
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 9 - альфа, цистеин-модифицированный
<400> 131
Met Ser Leu Ser Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu
1 5 10 15
Gly Pro Gly Ile Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe
20 25 30
Val Gln Glu Lys Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser
35 40 45
Asp Pro Ser Tyr Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu
50 55 60
Met Ile Phe Leu Ile Tyr Gln Gly Ser Tyr Asp Gln Gln Asn Ala Thr
65 70 75 80
Glu Gly Arg Tyr Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn
85 90 95
Leu Val Ile Ser Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys
100 105 110
Ala Met Arg Thr Ala Gly Gly Thr Ser Tyr Gly Lys Leu Thr Phe Gly
115 120 125
Gln Gly Thr Ile Leu Thr Val His Pro Asn Ile Gln Asn Pro Asp Pro
130 135 140
Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys
145 150 155 160
Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp
165 170 175
Ser Asp Val Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met
180 185 190
Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe
195 200 205
Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe
210 215 220
Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser
225 230 235 240
Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly
245 250 255
Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr
260 265 270
Leu Arg Leu Trp Ser Ser
275
<210> 132
<211> 311
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 9 - бета нативный
<400> 132
Met Ser Leu Gly Leu Leu Cys Cys Gly Ala Phe Ser Leu Leu Trp Ala
1 5 10 15
Gly Pro Val Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Arg Val Leu
20 25 30
Lys Thr Gly Gln Ser Met Thr Leu Leu Cys Ala Gln Asp Met Asn His
35 40 45
Glu Tyr Met Tyr Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu
50 55 60
Ile His Tyr Ser Val Gly Glu Gly Thr Thr Ala Lys Gly Glu Val Pro
65 70 75 80
Asp Gly Tyr Asn Val Ser Arg Leu Lys Lys Gln Asn Phe Leu Leu Gly
85 90 95
Leu Glu Ser Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser
100 105 110
Ser Tyr Phe Gly Thr Ala Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 133
<211> 311
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 9 - бета, цистеин-модифицированный
<400> 133
Met Ser Leu Gly Leu Leu Cys Cys Gly Ala Phe Ser Leu Leu Trp Ala
1 5 10 15
Gly Pro Val Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Arg Val Leu
20 25 30
Lys Thr Gly Gln Ser Met Thr Leu Leu Cys Ala Gln Asp Met Asn His
35 40 45
Glu Tyr Met Tyr Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu
50 55 60
Ile His Tyr Ser Val Gly Glu Gly Thr Thr Ala Lys Gly Glu Val Pro
65 70 75 80
Asp Gly Tyr Asn Val Ser Arg Leu Lys Lys Gln Asn Phe Leu Leu Gly
85 90 95
Leu Glu Ser Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser
100 105 110
Ser Tyr Phe Gly Thr Ala Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 134
<211> 280
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 10 - альфа нативный
<400> 134
Met Met Ile Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu
1 5 10 15
Ser Trp Val Trp Ser Gln Arg Lys Glu Val Glu Gln Asp Pro Gly Pro
20 25 30
Phe Asn Val Pro Glu Gly Ala Thr Val Ala Phe Asn Cys Thr Tyr Ser
35 40 45
Asn Ser Ala Ser Gln Ser Phe Phe Trp Tyr Arg Gln Asp Cys Arg Lys
50 55 60
Glu Pro Lys Leu Leu Met Ser Val Tyr Ser Ser Gly Asn Glu Asp Gly
65 70 75 80
Arg Phe Thr Ala Gln Leu Asn Arg Ala Ser Gln Tyr Ile Ser Leu Leu
85 90 95
Ile Arg Asp Ser Lys Leu Ser Asp Ser Ala Thr Tyr Leu Cys Val Val
100 105 110
Asn Phe Pro Ser Arg Gly Ala Gly Gly Thr Ser Tyr Gly Lys Leu Thr
115 120 125
Phe Gly Gln Gly Thr Ile Leu Thr Val His Pro Asn Ile Gln Lys Pro
130 135 140
Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser
145 150 155 160
Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser
165 170 175
Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg
180 185 190
Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser
195 200 205
Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Ala Asp
210 215 220
Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu
225 230 235 240
Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val
245 250 255
Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu
260 265 270
Met Thr Leu Arg Leu Trp Ser Ser
275 280
<210> 135
<211> 280
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 10 - альфа, цистеин-модифицированный
<400> 135
Met Met Ile Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu
1 5 10 15
Ser Trp Val Trp Ser Gln Arg Lys Glu Val Glu Gln Asp Pro Gly Pro
20 25 30
Phe Asn Val Pro Glu Gly Ala Thr Val Ala Phe Asn Cys Thr Tyr Ser
35 40 45
Asn Ser Ala Ser Gln Ser Phe Phe Trp Tyr Arg Gln Asp Cys Arg Lys
50 55 60
Glu Pro Lys Leu Leu Met Ser Val Tyr Ser Ser Gly Asn Glu Asp Gly
65 70 75 80
Arg Phe Thr Ala Gln Leu Asn Arg Ala Ser Gln Tyr Ile Ser Leu Leu
85 90 95
Ile Arg Asp Ser Lys Leu Ser Asp Ser Ala Thr Tyr Leu Cys Val Val
100 105 110
Asn Phe Pro Ser Arg Gly Ala Gly Gly Thr Ser Tyr Gly Lys Leu Thr
115 120 125
Phe Gly Gln Gly Thr Ile Leu Thr Val His Pro Asn Ile Gln Lys Pro
130 135 140
Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser
145 150 155 160
Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser
165 170 175
Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg
180 185 190
Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser
195 200 205
Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Ala Asp
210 215 220
Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu
225 230 235 240
Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val
245 250 255
Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu
260 265 270
Met Thr Leu Arg Leu Trp Ser Ser
275 280
<210> 136
<211> 7
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 3/ TCR 5/ TCR 15 / TCR 19 / TCR 21 / TCR 23 /
TCR 24 / TCR 25 / TCR 26 / TCR 29 - (E6)29 - TCR
альфа CDR1
<400> 136
Thr Ser Asp Gln Ser Tyr Gly
1 5
<210> 137
<211> 8
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 3/ TCR 5/ TCR 15 / TCR 19 / TCR 21 / TCR 23 /
TCR 25 / TCR 26 / TCR 29 - (E6)29 - TCR альфа
CDR2
<400> 137
Gln Gly Ser Tyr Asp Glu Gln Asn
1 5
<210> 138
<211> 11
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 3 альфа CDR3
<400> 138
Ala Met Arg Glu Gly Arg Gly Phe Lys Thr Ile
1 5 10
<210> 139
<211> 5
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 3 / TCR 13/ TCR 37/ TCR 53 бета CDR1
<400> 139
Ser Gly His Val Ser
1 5
<210> 140
<211> 6
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 3/ TCR 4/ TCR 13/ TCR 37 - (E6)29 бета CDR2
<400> 140
Phe Gln Asn Glu Ala Gln
1 5
<210> 141
<211> 13
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 3 бета CDR3
<400> 141
Ala Ser Ser His Leu Ala Gly Phe Thr Gly Glu Leu Phe
1 5 10
<210> 142
<211> 7
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 4/ TCR 27 - (E6)29/ TCR 11
альфа CDR1
<400> 142
Thr Ile Ser Gly Thr Asp Tyr
1 5
<210> 143
<211> 5
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 4/ TCR 27 - (E6)29/ TCR 11 альфа CDR2
<400> 143
Gly Leu Thr Ser Asn
1 5
<210> 144
<211> 14
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 4 - (E6)29 альфа CDR3
<400> 144
Ile Leu Leu Val Ile Arg Gly Thr Ser Tyr Gly Lys Leu Thr
1 5 10
<210> 145
<211> 5
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 4 - (E6)29 бета CDR1
<400> 145
Ser Glu His Asn Arg
1 5
<210> 146
<211> 12
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 4 - (E6)29 бета CDR3
<400> 146
Ala Ser Ser Pro Gly Gly Gly Asn Thr Glu Ala Phe
1 5 10
<210> 147
<211> 14
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 5 - (E6)29 - TCR альфа CDR3
<400> 147
Ala Met Arg Glu Gly Thr Gly Thr Ser Tyr Gly Lys Leu Thr
1 5 10
<210> 148
<211> 5
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 5/ TCR 16/ TCR 17 /TCR 18 /TCR 19 / TCR 23
/TCR 24 /TCR 25 /TCR 28 - (E6)29/ TCR 8 / TCR 10
/ TCR 14 - TCR бета CDR1
<400> 148
Met Asp His Glu Asn
1 5
<210> 149
<211> 6
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 5/ TCR 16/ TCR 17/ TCR 18/ TCR 19 / TCR 23/
TCR 24/ TCR 25/ TCR 28- (E6)29 / TCR 8 / TCR 10 /
TCR 14 - TCR бета CDR2
<400> 149
Ser Tyr Asp Val Lys Met
1 5
<210> 150
<211> 13
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 5 - (E6)29 - TCR бета CDR3
<400> 150
Ala Ser Ser Pro Trp Gly Glu Thr His Gln Pro Gln His
1 5 10
<210> 151
<211> 6
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 6, TCR 12, TCR 50, TCR 55 альфа CDR1
<400> 151
Asp Ser Ser Ser Thr Tyr
1 5
<210> 152
<211> 7
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 6, TCR 12, TCR 50, TCR 55 альфа CDR2
<400> 152
Ile Phe Ser Asn Met Asp Met
1 5
<210> 153
<211> 12
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 6 альфа CDR3
<400> 153
Ala Glu Ser Ile Arg Gly Phe Gly Asn Val Leu His
1 5 10
<210> 154
<211> 5
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 6/ TCR 7 - (E7)11, E7(11-19) / TCR 12
консенсус, TCR 30 / TCR 33 / TCR 36 / TCR 39 /TCR
40 / TCR 41 / TCR 42 / TCR 43 / TCR 47 / TCR 48 /
TCR 49 / TCR 51 / TCR 54/ TCR 55/TCR 66 бета CDR1
<400> 154
Ser Asn His Leu Tyr
1 5
<210> 155
<211> 6
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 6/ TCR 7 - (E7)11, E7(11-19) / TCR 12
консенсус, TCR 30 / TCR 33 / TCR 36 / TCR 39 / TCR
42 / TCR 43 / TCR 47 / TCR 48 / TCR 49 / TCR 51 /
TCR 54/ TCR 55/TCR 66 бета CDR2
<400> 155
Phe Tyr Asn Asn Glu Ile
1 5
<210> 156
<211> 11
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 6 / TCR 12/ TCR 55 бета CDR3
<400> 156
Ala Ser Thr Thr Arg Ser Ser Tyr Glu Gln Tyr
1 5 10
<210> 157
<211> 6
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 7/ TCR 54 альфа CDR1
<400> 157
Val Ser Pro Phe Ser Asn
1 5
<210> 158
<211> 7
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 7/ TCR 54 - (E7)11 - альфа CDR2
<400> 158
Met Thr Phe Ser Glu Asn Thr
1 5
<210> 159
<211> 12
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 7/ TCR 54 - (E7)11 - альфа CDR3
<400> 159
Val Val Ser Arg Asp Asn Tyr Gly Gln Asn Phe Val
1 5 10
<210> 160
<211> 11
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 7/ TCR 54 - (E7)11 - бета CDR3
<400> 160
Ala Ile Thr Asp Arg Thr Asn Tyr Gly Tyr Thr
1 5 10
<210> 161
<211> 6
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 8/ TCR 16 / TCR 18 альфа CDR1
<400> 161
Asp Ser Ala Ile Tyr Asn
1 5
<210> 162
<211> 7
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 8/ TCR 16 / TCR 18 альфа CDR2
<400> 162
Ile Gln Ser Ser Gln Arg Glu
1 5
<210> 163
<211> 11
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 8 альфа CDR3
<400> 163
Ala Val Arg Pro Leu Gly Asn Thr Pro Leu Val
1 5 10
<210> 164
<211> 13
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 8 бета CDR3
<400> 164
Ala Ser Ser Leu Trp Gly Ala Ser Thr Asp Thr Gln Tyr
1 5 10
<210> 165
<211> 7
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 9 / TCR 17 альфа CDR1
<400> 165
Thr Ser Asp Pro Ser Tyr Gly
1 5
<210> 166
<211> 8
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 9 / TCR 17 альфа CDR2
<400> 166
Gln Gly Ser Tyr Asp Gln Gln Asn
1 5
<210> 167
<211> 14
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 9 альфа CDR3
<400> 167
Ala Met Arg Thr Ala Gly Gly Thr Ser Tyr Gly Lys Leu Thr
1 5 10
<210> 168
<211> 5
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 9/ TCR 26 бета CDR1
<400> 168
Met Asn His Glu Tyr
1 5
<210> 169
<211> 6
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 9/ TCR 26 бета CDR2
<400> 169
Ser Val Gly Glu Gly Thr
1 5
<210> 170
<211> 12
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 9 бета CDR3
<400> 170
Ala Ser Ser Tyr Phe Gly Thr Ala Tyr Glu Gln Tyr
1 5 10
<210> 171
<211> 6
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 10/ TCR 28/ TCR 36/ TCR 41/ TCR 66 альфа CDR1
<400> 171
Asn Ser Ala Ser Gln Ser
1 5
<210> 172
<211> 6
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 10/ TCR 28/ TCR 41/ TCR 66 альфа CDR2
<400> 172
Val Tyr Ser Ser Gly Asn
1 5
<210> 173
<211> 18
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 10 альфа CDR3
<400> 173
Val Val Asn Phe Pro Ser Arg Gly Ala Gly Gly Thr Ser Tyr Gly Lys
1 5 10 15
Leu Thr
<210> 174
<211> 13
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 10 бета CDR3
<400> 174
Ala Ser Ser Leu Ser Leu Thr Gly Asn Tyr Gly Tyr Thr
1 5 10
<210> 175
<211> 13
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 11 альфа CDR3
<400> 175
Ile Leu Ser Ala His Ser Asn Ser Gly Tyr Ala Leu Asn
1 5 10
<210> 176
<211> 5
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 11 бета CDR1
<400> 176
Ser Gln Val Thr Met
1 5
<210> 177
<211> 7
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 11 бета CDR2
<400> 177
Ala Asn Gln Gly Ser Glu Ala
1 5
<210> 178
<211> 15
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 11 бета CDR3
<400> 178
Ser Val Val Pro Trp Thr Arg Gly Gly Ser Thr Asp Thr Gln Tyr
1 5 10 15
<210> 179
<211> 310
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 10 - бета нативный
<400> 179
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Ser Leu Ser Leu Thr Gly Asn Tyr Gly Tyr Thr Phe Gly Ser Gly Thr
115 120 125
Arg Leu Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe
305 310
<210> 180
<211> 310
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 10 - бета, цистеин-модифицированный
<400> 180
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Ser Leu Ser Leu Thr Gly Asn Tyr Gly Tyr Thr Phe Gly Ser Gly Thr
115 120 125
Arg Leu Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe
305 310
<210> 181
<211> 20
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 3/ TCR 9/ TCR 5/ TCR 15/ TCR 17/ TCR 19/ TCR
21/ TCR 23/ TCR 24/ TCR 25/ TCR 26/ TCR 29 -
(E6)29 TCR альфа, Сигнальный пептид
<400> 181
Met Ser Leu Ser Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu
1 5 10 15
Gly Pro Gly Ile
20
<210> 182
<211> 19
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 3 / TCR 13/ TCR 37 - бета, Сигнальный пептид
<400> 182
Met Gly Thr Arg Leu Leu Cys Trp Val Val Leu Gly Phe Leu Gly Thr
1 5 10 15
Asp His Thr
<210> 183
<211> 810
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 12 - альфа нативный
<400> 183
atgaagacat ttgctggatt ttcgttcctg tttttgtggc tgcagctgga ctgtatgagt 60
agaggagagg atgtggagca gagtcttttc ctgagtgtcc gagagggaga cagctccgtt 120
ataaactgca cttacacaga cagctcctcc acctacttat actggtataa gcaagaacct 180
ggagcaggtc tccagttgct gacgtatatt ttttcaaata tggacatgaa acaagaccaa 240
agactcactg ttctattgaa taaaaaggat aaacatctgt ctctgcgcat tgcagacacc 300
cagactgggg actcagctat ctacttctgt gcagtcccct cgggtgctac aaacaagctc 360
atctttggaa ctggcactct gcttgctgtc cagccaaata tccagaaccc tgaccctgcc 420
gtgtaccagc tgagagactc taaatccagt gacaagtctg tctgcctatt caccgatttt 480
gattctcaaa caaatgtgtc acaaagtaag gattctgatg tgtatatcac agacaaatgc 540
gtgctagaca tgaggtctat ggacttcaag agcaacagtg ctgtggcctg gagcaacaaa 600
tctgactttg catgtgcaaa cgccttcaac aacagcatta ttccagaaga caccttcttc 660
cccagcccag aaagttcctg tgatgtcaag ctggtcgaga aaagctttga aacagatacg 720
aacctaaact ttcaaaacct gtcagtgatt gggttccgaa tcctcctcct gaaagtggcc 780
gggtttaatc tgctcatgac gctgcggctg 810
<210> 184
<211> 17
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 4/ TCR 27 - (E6)29 альфа, Сигнальный пептид
<400> 184
Met Lys Leu Val Thr Ser Ile Thr Val Leu Leu Ser Leu Gly Ile Met
1 5 10 15
Gly
<210> 185
<211> 21
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 4 - (E6)29 бета, Сигнальный пептид
<400> 185
Met Gly Thr Ser Leu Leu Cys Trp Met Ala Leu Cys Leu Leu Gly Ala
1 5 10 15
Asp His Ala Asp Thr
20
<210> 186
<211> 19
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 5/ TCR 16/ TCR 17 / TCR 18/ TCR 19/ TCR 23/
TCR 24/ TCR 25/ TCR 28 - (E6)29/ TCR 8/ TCR10 /
TCR 14 - TCR бета, Сигнальный пептид
<400> 186
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val
<210> 187
<211> 21
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 6 / TCR 12/ TCR 50/ TCR 55- альфа, Сигнальный
пептид
<400> 187
Met Lys Thr Phe Ala Gly Phe Ser Phe Leu Phe Leu Trp Leu Gln Leu
1 5 10 15
Asp Cys Met Ser Arg
20
<210> 188
<211> 19
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 6/7/12/33/36/39/43/47/49/51/54/55/30/66 (E6)29
- бета, Сигнальный пептид
<400> 188
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr
<210> 189
<211> 21
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 7/ TCR 54 - (E7)11 - альфа, Сигнальный пептид
<400> 189
Met Lys Lys His Leu Thr Thr Phe Leu Val Ile Leu Trp Leu Tyr Phe
1 5 10 15
Tyr Arg Gly Asn Gly
20
<210> 190
<211> 19
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 8/ TCR 16 / TCR 18 - альфа, Сигнальный пептид
<400> 190
Met Glu Thr Leu Leu Gly Leu Leu Ile Leu Trp Leu Gln Leu Gln Trp
1 5 10 15
Val Ser Ser
<210> 191
<211> 19
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 9/ TCR 26 - бета, Сигнальный пептид
<400> 191
Met Ser Leu Gly Leu Leu Cys Cys Gly Ala Phe Ser Leu Leu Trp Ala
1 5 10 15
Gly Pro Val
<210> 192
<211> 22
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 10/ TCR 28 / TCR 36 / TCR 41/TCR 66 - альфа
Сигнальный пептид
<400> 192
Met Met Ile Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu
1 5 10 15
Ser Trp Val Trp Ser Gln
20
<210> 193
<211> 17
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 11 - альфа, Сигнальный пептид
<400> 193
Met Lys Leu Val Thr Ser Ile Thr Val Leu Leu Ser Leu Gly Ile Met
1 5 10 15
Gly
<210> 194
<211> 15
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 11 - бета, Сигнальный пептид
<400> 194
Met Leu Ser Leu Leu Leu Leu Leu Leu Gly Leu Gly Ser Val Phe
1 5 10 15
<210> 195
<211> 270
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 11 - альфа нативный
<400> 195
Met Lys Leu Val Thr Ser Ile Thr Val Leu Leu Ser Leu Gly Ile Met
1 5 10 15
Gly Asp Ala Lys Thr Thr Gln Pro Asn Ser Met Glu Ser Asn Glu Glu
20 25 30
Glu Pro Val His Leu Pro Cys Asn His Ser Thr Ile Ser Gly Thr Asp
35 40 45
Tyr Ile His Trp Tyr Arg Gln Leu Pro Ser Gln Gly Pro Glu Tyr Val
50 55 60
Ile His Gly Leu Thr Ser Asn Val Asn Asn Arg Met Ala Ser Leu Ala
65 70 75 80
Ile Ala Glu Asp Arg Lys Ser Ser Thr Leu Ile Leu His Arg Ala Thr
85 90 95
Leu Arg Asp Ala Ala Val Tyr Tyr Cys Ile Leu Ser Ala His Ser Asn
100 105 110
Ser Gly Tyr Ala Leu Asn Phe Gly Lys Gly Thr Ser Leu Leu Val Thr
115 120 125
Pro His Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser
130 135 140
Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln
145 150 155 160
Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys
165 170 175
Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val
180 185 190
Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn
195 200 205
Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys
210 215 220
Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn
225 230 235 240
Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val
245 250 255
Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
260 265 270
<210> 196
<211> 141
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 4 - (E6)29 / TCR 5 - (E6)29 / TCR 12/ TCR 55 -
TCR альфа, Константная область
<400> 196
Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys
1 5 10 15
Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr
20 25 30
Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys
35 40 45
Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala
50 55 60
Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser
65 70 75 80
Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp
85 90 95
Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe
100 105 110
Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala
115 120 125
Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
130 135 140
<210> 197
<211> 177
<212> Белок
<213> Homo sapiens
<220>
<223> TCR
4/5/7/10/14/16/17/18/21/22/23/25/27/28/30/37/39/50
/54 - TCR бета, Константная область
<400> 197
Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val Phe Glu Pro
1 5 10 15
Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys Leu
20 25 30
Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp Trp Val Asn
35 40 45
Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro Leu Lys
50 55 60
Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg Leu
65 70 75 80
Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg Cys
85 90 95
Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln Asp
100 105 110
Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly Arg
115 120 125
Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly Val Leu Ser
130 135 140
Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr Ala
145 150 155 160
Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys Asp
165 170 175
Phe
<210> 198
<211> 141
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 3/ TCR 10 TCR альфа, Константная область
<400> 198
Asn Ile Gln Lys Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys
1 5 10 15
Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr
20 25 30
Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys
35 40 45
Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala
50 55 60
Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser
65 70 75 80
Ile Ile Pro Ala Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp
85 90 95
Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe
100 105 110
Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala
115 120 125
Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
130 135 140
<210> 199
<211> 179
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 3/ 6/8/9/11/13/19/ 20/24/29/31/32/33/34/
35/36/38/ 40/41/42/43/ 45/46/47/48/49/51/52/55/66
- TCR бета, Константная область
<400> 199
Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu Pro
1 5 10 15
Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys Leu
20 25 30
Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val Asn
35 40 45
Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro Leu Lys
50 55 60
Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg Leu
65 70 75 80
Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg Cys
85 90 95
Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln Asp
100 105 110
Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly Arg
115 120 125
Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu Ser
130 135 140
Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr Ala
145 150 155 160
Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys Asp
165 170 175
Ser Arg Gly
<210> 200
<211> 141
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 6/ TCR 11 альфа, Константная область
<400> 200
His Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys
1 5 10 15
Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr
20 25 30
Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys
35 40 45
Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala
50 55 60
Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser
65 70 75 80
Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp
85 90 95
Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe
100 105 110
Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala
115 120 125
Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
130 135 140
<210> 201
<211> 141
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 7/ TCR 14/ TCR 15 / TCR 20/ TCR 36/ TCR 54/TCR
66 - альфа, Константная область
<400> 201
Tyr Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys
1 5 10 15
Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr
20 25 30
Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys
35 40 45
Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala
50 55 60
Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser
65 70 75 80
Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp
85 90 95
Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe
100 105 110
Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala
115 120 125
Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
130 135 140
<210> 202
<211> 801
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 13 - альфа нативный
<400> 202
atgctcctgc tgctcgtccc agtgctcgag gtgattttta ctctgggagg aaccagagcc 60
cagtcggtga cccagcttga cagccacgtc tctgtctctg aaggaacccc ggtgctgctg 120
aggtgcaact actcatcttc ttattcacca tctctcttct ggtatgtgca acaccccaac 180
aaaggactcc agcttctcct gaagtacaca tcagcggcca ccctggttaa aggcatcaac 240
ggttttgagg ctgaatttaa gaagagtgaa acctccttcc acctgacgaa accctcagcc 300
catatgagcg acgcggctga gtacttctgt gttgtgaggg gaggaaagct tatcttcgga 360
cagggaacgg agttatctgt gaaacccaat atccagaacc ctgaccctgc cgtgtaccag 420
ctgagagact ctaaatccag tgacaagtct gtctgcctat tcaccgattt tgattctcaa 480
acaaatgtgt cacaaagtaa ggattctgat gtgtatatca cagacaaatg cgtgctagac 540
atgaggtcta tggacttcaa gagcaacagt gctgtggcct ggagcaacaa atctgacttt 600
gcatgtgcaa acgccttcaa caacagcatt attccagaag acaccttctt ccccagccca 660
gaaagttcct gtgatgtcaa gctggtcgag aaaagctttg aaacagatac gaacctaaac 720
tttcaaaacc tgtcagtgat tgggttccga atcctcctcc tgaaagtggc cgggtttaat 780
ctgctcatga cgctgcggct g 801
<210> 203
<211> 141
<212> Белок
<213> Homo sapiens
<220>
<223> TCR
8/9/13/16/17/18/21/26/27/28/30/31/32/33/34/35/37/3
8/39/40/41/42/43/44/45/46/48/49/50/51/52/53 -
альфа, Константная область
<400> 203
Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys
1 5 10 15
Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr
20 25 30
Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys
35 40 45
Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala
50 55 60
Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser
65 70 75 80
Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp
85 90 95
Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe
100 105 110
Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala
115 120 125
Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
130 135 140
<210> 204
<211> 22
<212> Белок
<213> Искусственная последовательность
<220>
<223> P2A
<400> 204
Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu Lys Gln Ala Gly Asp Val
1 5 10 15
Glu Glu Asn Pro Gly Pro
20
<210> 205
<211> 270
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 11 - альфа, цистеин-модифицированный
<400> 205
Met Lys Leu Val Thr Ser Ile Thr Val Leu Leu Ser Leu Gly Ile Met
1 5 10 15
Gly Asp Ala Lys Thr Thr Gln Pro Asn Ser Met Glu Ser Asn Glu Glu
20 25 30
Glu Pro Val His Leu Pro Cys Asn His Ser Thr Ile Ser Gly Thr Asp
35 40 45
Tyr Ile His Trp Tyr Arg Gln Leu Pro Ser Gln Gly Pro Glu Tyr Val
50 55 60
Ile His Gly Leu Thr Ser Asn Val Asn Asn Arg Met Ala Ser Leu Ala
65 70 75 80
Ile Ala Glu Asp Arg Lys Ser Ser Thr Leu Ile Leu His Arg Ala Thr
85 90 95
Leu Arg Asp Ala Ala Val Tyr Tyr Cys Ile Leu Ser Ala His Ser Asn
100 105 110
Ser Gly Tyr Ala Leu Asn Phe Gly Lys Gly Thr Ser Leu Leu Val Thr
115 120 125
Pro His Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser
130 135 140
Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln
145 150 155 160
Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys
165 170 175
Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val
180 185 190
Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn
195 200 205
Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys
210 215 220
Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn
225 230 235 240
Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val
245 250 255
Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
260 265 270
<210> 206
<211> 312
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 11 - бета нативный
<400> 206
Met Leu Ser Leu Leu Leu Leu Leu Leu Gly Leu Gly Ser Val Phe Ser
1 5 10 15
Ala Val Ile Ser Gln Lys Pro Ser Arg Asp Ile Cys Gln Arg Gly Thr
20 25 30
Ser Leu Thr Ile Gln Cys Gln Val Asp Ser Gln Val Thr Met Met Phe
35 40 45
Trp Tyr Arg Gln Gln Pro Gly Gln Ser Leu Thr Leu Ile Ala Thr Ala
50 55 60
Asn Gln Gly Ser Glu Ala Thr Tyr Glu Ser Gly Phe Val Ile Asp Lys
65 70 75 80
Phe Pro Ile Ser Arg Pro Asn Leu Thr Phe Ser Thr Leu Thr Val Ser
85 90 95
Asn Met Ser Pro Glu Asp Ser Ser Ile Tyr Leu Cys Ser Val Val Pro
100 105 110
Trp Thr Arg Gly Gly Ser Thr Asp Thr Gln Tyr Phe Gly Pro Gly Thr
115 120 125
Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 207
<211> 66
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCR 3/ TCR 6/ TCR 8/ TCR 9/ TCR 10/TCR 11/ P2A
<400> 207
ggatccggag ctaccaactt ctctctgctg aaacaggcag gcgatgtgga ggaaaatcct 60
gggcca 66
<210> 208
<211> 66
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCR 4/ P2A
<400> 208
gggagtggag caacaaactt ttcactgctg aagcaggccg gcgatgtgga ggaaaatcct 60
gggcca 66
<210> 209
<211> 66
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCR 5/ P2A
<400> 209
gggtccggag ccacaaattt ttctctgctg aaacaggctg gcgatgtgga ggaaaaccct 60
gggcca 66
<210> 210
<211> 66
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCR 7/ P2A
<400> 210
ggaagcggcg caacaaactt ttccctgctg aaacaggccg gagatgtgga ggaaaatcct 60
ggccca 66
<210> 211
<211> 18
<212> Белок
<213> Искусственная последовательность
<220>
<223> T2A
<400> 211
Glu Gly Arg Gly Ser Leu Leu Thr Cys Gly Asp Val Glu Glu Asn Pro
1 5 10 15
Gly Pro
<210> 212
<211> 141
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 3/ TCR 10 нативный TCR альфа, константная область
<400> 212
Asn Ile Gln Lys Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys
1 5 10 15
Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr
20 25 30
Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr
35 40 45
Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala
50 55 60
Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser
65 70 75 80
Ile Ile Pro Ala Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp
85 90 95
Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe
100 105 110
Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala
115 120 125
Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
130 135 140
<210> 213
<211> 141
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 4/5/12/8/9/13/16/17/18
/21/26/27/28/30/31/32/33/34/35/37/38/39/40
/41/42/43/44/45/46/48/49/50/51/52/53/55 - нативный
TCR альфа, Константная область
<400> 213
Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys
1 5 10 15
Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr
20 25 30
Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr
35 40 45
Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala
50 55 60
Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser
65 70 75 80
Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp
85 90 95
Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe
100 105 110
Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala
115 120 125
Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
130 135 140
<210> 214
<211> 177
<212> Белок
<213> Homo sapiens
<220>
<223> TCR
4/5/16/17/18/21/22/23/25/27/28/7/37/39/50/51/52/54
/10/14 - нативный TCR бета, Константная область
<400> 214
Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val Phe Glu Pro
1 5 10 15
Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys Leu
20 25 30
Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp Trp Val Asn
35 40 45
Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro Leu Lys
50 55 60
Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg Leu
65 70 75 80
Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg Cys
85 90 95
Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln Asp
100 105 110
Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly Arg
115 120 125
Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly Val Leu Ser
130 135 140
Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr Ala
145 150 155 160
Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys Asp
165 170 175
Phe
<210> 215
<211> 142
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 3/ TCR 10 нативный TCR альфа, Константная область
<400> 215
Pro Asn Ile Gln Lys Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser
1 5 10 15
Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln
20 25 30
Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys
35 40 45
Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val
50 55 60
Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn
65 70 75 80
Ser Ile Ile Pro Ala Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys
85 90 95
Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn
100 105 110
Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val
115 120 125
Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
130 135 140
<210> 216
<211> 179
<212> Белок
<213> Homo sapiens
<220>
<223> TCR
3/6/12/8/9/11/13/19/20/24/29/31/32/33/34/35/36/38/
40/41/42/43/46/47/48/49/53/55/66
нативный TCR бета, Константная область
<400> 216
Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu Pro
1 5 10 15
Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys Leu
20 25 30
Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val Asn
35 40 45
Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro Leu Lys
50 55 60
Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg Leu
65 70 75 80
Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg Cys
85 90 95
Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln Asp
100 105 110
Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly Arg
115 120 125
Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu Ser
130 135 140
Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr Ala
145 150 155 160
Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys Asp
165 170 175
Ser Arg Gly
<210> 217
<211> 141
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 6/ TCR 11 нативный TCR альфа, Константная область
<400> 217
His Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys
1 5 10 15
Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr
20 25 30
Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr
35 40 45
Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala
50 55 60
Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser
65 70 75 80
Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp
85 90 95
Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe
100 105 110
Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala
115 120 125
Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
130 135 140
<210> 218
<211> 141
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 7/ TCR 14/ TCR 15/ TCR 20/ TCR 36/ TCR 54/TCR
66 - нативный TCR альфа, Константная область
<400> 218
Tyr Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys
1 5 10 15
Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr
20 25 30
Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr
35 40 45
Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala
50 55 60
Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser
65 70 75 80
Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp
85 90 95
Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe
100 105 110
Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala
115 120 125
Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
130 135 140
<210> 219
<211> 813
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 14 - альфа нативный
<400> 219
atggagaaga atcctttggc agccccatta ctaatcctct ggtttcatct tgactgcgtg 60
agcagcatac tgaacgtgga acaaagtcct cagtcactgc atgttcagga gggagacagc 120
accaatttca cctgcagctt cccttccagc aatttttatg ccttacactg gtacagatgg 180
gaaactgcaa aaagccccga ggccttgttt gtaatgactt taaatgggga tgaaaagaag 240
aaaggacgaa taagtgccac tcttaatacc aaggagggtt acagctattt gtacatcaaa 300
ggatcccagc ctgaagactc agccacatac ctctgtgcct ctcaaactgg ggcaaacaac 360
ctcttctttg ggactggaac gagactcacc gttattccct atatccagaa ccctgaccct 420
gccgtgtacc agctgagaga ctctaaatcc agtgacaagt ctgtctgcct attcaccgat 480
tttgattctc aaacaaatgt gtcacaaagt aaggattctg atgtgtatat cacagacaaa 540
actgtgctag acatgaggtc tatggacttc aagagcaaca gtgctgtggc ctggagcaac 600
aaatctgact ttgcatgtgc aaacgccttc aacaacagca ttattccaga agacaccttc 660
ttccccagcc cagaaagttc ctgtgatgtc aagctggtcg agaaaagctt tgaaacagat 720
acgaacctaa actttcaaaa cctgtcagtg attgggttcc gaatcctcct cctgaaagtg 780
gccgggttta atctgctcat gacgctgcgg ctg 813
<210> 220
<211> 142
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 4 - (E6)29 / TCR 5 / TCR 12 /TCR 8/ TCR 9 /
TCR 13- (E6)29 - нативный TCR альфа, Константная область
<400> 220
Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser
1 5 10 15
Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln
20 25 30
Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys
35 40 45
Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val
50 55 60
Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn
65 70 75 80
Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys
85 90 95
Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn
100 105 110
Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val
115 120 125
Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
130 135 140
<210> 221
<211> 312
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 11 - бета, цистеин-модифицированный
<400> 221
Met Leu Ser Leu Leu Leu Leu Leu Leu Gly Leu Gly Ser Val Phe Ser
1 5 10 15
Ala Val Ile Ser Gln Lys Pro Ser Arg Asp Ile Cys Gln Arg Gly Thr
20 25 30
Ser Leu Thr Ile Gln Cys Gln Val Asp Ser Gln Val Thr Met Met Phe
35 40 45
Trp Tyr Arg Gln Gln Pro Gly Gln Ser Leu Thr Leu Ile Ala Thr Ala
50 55 60
Asn Gln Gly Ser Glu Ala Thr Tyr Glu Ser Gly Phe Val Ile Asp Lys
65 70 75 80
Phe Pro Ile Ser Arg Pro Asn Leu Thr Phe Ser Thr Leu Thr Val Ser
85 90 95
Asn Met Ser Pro Glu Asp Ser Ser Ile Tyr Leu Cys Ser Val Val Pro
100 105 110
Trp Thr Arg Gly Gly Ser Thr Asp Thr Gln Tyr Phe Gly Pro Gly Thr
115 120 125
Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 222
<211> 273
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 12/ TCR 55 - (E7)11 - альфа нативный
<400> 222
Met Lys Thr Phe Ala Gly Phe Ser Phe Leu Phe Leu Trp Leu Gln Leu
1 5 10 15
Asp Cys Met Ser Arg Gly Glu Asp Val Glu Gln Ser Leu Phe Leu Ser
20 25 30
Val Arg Glu Gly Asp Ser Ser Val Ile Asn Cys Thr Tyr Thr Asp Ser
35 40 45
Ser Ser Thr Tyr Leu Tyr Trp Tyr Lys Gln Glu Pro Gly Ala Gly Leu
50 55 60
Gln Leu Leu Thr Tyr Ile Phe Ser Asn Met Asp Met Lys Gln Asp Gln
65 70 75 80
Arg Leu Thr Val Leu Leu Asn Lys Lys Asp Lys His Leu Ser Leu Arg
85 90 95
Ile Ala Asp Thr Gln Thr Gly Asp Ser Ala Ile Tyr Phe Cys Ala Val
100 105 110
Pro Ser Gly Ala Thr Asn Lys Leu Ile Phe Gly Thr Gly Thr Leu Leu
115 120 125
Ala Val Gln Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu
130 135 140
Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe
145 150 155 160
Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile
165 170 175
Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn
180 185 190
Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala
195 200 205
Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu
210 215 220
Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr
225 230 235 240
Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu
245 250 255
Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser
260 265 270
Ser
<210> 223
<211> 610
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 3 полноразмерная последовательность, нативный
<400> 223
Met Gly Thr Arg Leu Leu Cys Trp Val Val Leu Gly Phe Leu Gly Thr
1 5 10 15
Asp His Thr Gly Ala Gly Val Ser Gln Ser Pro Arg Tyr Lys Val Ala
20 25 30
Lys Arg Gly Gln Asp Val Ala Leu Arg Cys Asp Pro Ile Ser Gly His
35 40 45
Val Ser Leu Phe Trp Tyr Gln Gln Ala Leu Gly Gln Gly Pro Glu Phe
50 55 60
Leu Thr Tyr Phe Gln Asn Glu Ala Gln Leu Asp Lys Ser Gly Leu Pro
65 70 75 80
Ser Asp Arg Phe Phe Ala Glu Arg Pro Glu Gly Ser Val Ser Thr Leu
85 90 95
Lys Ile Gln Arg Thr Gln Gln Glu Asp Ser Ala Val Tyr Leu Cys Ala
100 105 110
Ser Ser His Leu Ala Gly Phe Thr Gly Glu Leu Phe Phe Gly Glu Gly
115 120 125
Ser Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu
130 135 140
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
145 150 155 160
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu
165 170 175
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr
180 185 190
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
195 200 205
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
210 215 220
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
225 230 235 240
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
245 250 255
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr
260 265 270
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
275 280 285
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
290 295 300
Met Val Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe
305 310 315 320
Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met
325 330 335
Ser Leu Ser Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu Gly
340 345 350
Pro Gly Ile Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val
355 360 365
Gln Glu Lys Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp
370 375 380
Gln Ser Tyr Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met
385 390 395 400
Ile Phe Leu Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu
405 410 415
Gly Arg Tyr Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu
420 425 430
Val Ile Ser Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala
435 440 445
Met Arg Glu Gly Arg Gly Phe Lys Thr Ile Phe Gly Ala Gly Thr Arg
450 455 460
Leu Phe Val Lys Ala Asn Ile Gln Lys Pro Asp Pro Ala Val Tyr Gln
465 470 475 480
Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp
485 490 495
Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr
500 505 510
Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser
515 520 525
Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn
530 535 540
Ala Phe Asn Asn Ser Ile Ile Pro Ala Asp Thr Phe Phe Pro Ser Pro
545 550 555 560
Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp
565 570 575
Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu
580 585 590
Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp
595 600 605
Ser Ser
610
<210> 224
<211> 603
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 4 - (E6)29 полноразмерная последовательность
нативный
<400> 224
Met Gly Thr Ser Leu Leu Cys Trp Met Ala Leu Cys Leu Leu Gly Ala
1 5 10 15
Asp His Ala Asp Thr Gly Val Ser Gln Asp Pro Arg His Lys Ile Thr
20 25 30
Lys Arg Gly Gln Asn Val Thr Phe Arg Cys Asp Pro Ile Ser Glu His
35 40 45
Asn Arg Leu Tyr Trp Tyr Arg Gln Thr Leu Gly Gln Gly Pro Glu Phe
50 55 60
Leu Thr Tyr Phe Gln Asn Glu Ala Gln Leu Glu Lys Ser Arg Leu Leu
65 70 75 80
Ser Asp Arg Phe Ser Ala Glu Arg Pro Lys Gly Ser Phe Ser Thr Leu
85 90 95
Glu Ile Gln Arg Thr Glu Gln Gly Asp Ser Ala Met Tyr Leu Cys Ala
100 105 110
Ser Ser Pro Gly Gly Gly Asn Thr Glu Ala Phe Phe Gly Gln Gly Thr
115 120 125
Arg Leu Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu
305 310 315 320
Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Lys Leu Val
325 330 335
Thr Ser Ile Thr Val Leu Leu Ser Leu Gly Ile Met Gly Asp Ala Lys
340 345 350
Thr Thr Gln Pro Asn Ser Met Glu Ser Asn Glu Glu Glu Pro Val His
355 360 365
Leu Pro Cys Asn His Ser Thr Ile Ser Gly Thr Asp Tyr Ile His Trp
370 375 380
Tyr Arg Gln Leu Pro Ser Gln Gly Pro Glu Tyr Val Ile His Gly Leu
385 390 395 400
Thr Ser Asn Val Asn Asn Arg Met Ala Ser Leu Ala Ile Ala Glu Asp
405 410 415
Arg Lys Ser Ser Thr Leu Ile Leu His Arg Ala Thr Leu Arg Asp Ala
420 425 430
Ala Val Tyr Tyr Cys Ile Leu Leu Val Ile Arg Gly Thr Ser Tyr Gly
435 440 445
Lys Leu Thr Phe Gly Gln Gly Thr Ile Leu Thr Val His Pro Asn Ile
450 455 460
Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser
465 470 475 480
Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val
485 490 495
Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr Val Leu
500 505 510
Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser
515 520 525
Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile
530 535 540
Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys
545 550 555 560
Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn
565 570 575
Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe
580 585 590
Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600
<210> 225
<211> 610
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 5 - (E6)29 - TCR полноразмерная последовательность
нативный
<400> 225
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Ser Pro Trp Gly Glu Thr His Gln Pro Gln His Phe Gly Asp Gly Thr
115 120 125
Arg Leu Ser Ile Leu Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu
305 310 315 320
Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Ser Leu Ser
325 330 335
Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu Gly Pro Gly Ile
340 345 350
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
355 360 365
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln Ser Tyr
370 375 380
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
385 390 395 400
Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly Arg Tyr
405 410 415
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
420 425 430
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Arg Glu
435 440 445
Gly Thr Gly Thr Ser Tyr Gly Lys Leu Thr Phe Gly Gln Gly Thr Ile
450 455 460
Leu Thr Val His Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln
465 470 475 480
Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp
485 490 495
Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr
500 505 510
Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser
515 520 525
Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn
530 535 540
Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro
545 550 555 560
Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp
565 570 575
Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu
580 585 590
Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp
595 600 605
Ser Ser
610
<210> 226
<211> 607
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 6 полноразмерная последовательность, нативный
<400> 226
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ser Thr Thr Arg Ser Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser Leu
305 310 315 320
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Lys Thr
325 330 335
Phe Ala Gly Phe Ser Phe Leu Phe Leu Trp Leu Gln Leu Asp Cys Met
340 345 350
Ser Arg Gly Glu Asp Val Glu Gln Ser Leu Phe Leu Ser Val Arg Glu
355 360 365
Gly Asp Ser Ser Val Ile Asn Cys Thr Tyr Thr Asp Ser Ser Ser Thr
370 375 380
Tyr Leu Tyr Trp Tyr Lys Gln Glu Pro Gly Ala Gly Leu Gln Leu Leu
385 390 395 400
Thr Tyr Ile Phe Ser Asn Met Asp Met Lys Gln Asp Gln Arg Leu Thr
405 410 415
Val Leu Leu Asn Lys Lys Asp Lys His Leu Ser Leu Arg Ile Ala Asp
420 425 430
Thr Gln Thr Gly Asp Ser Ala Ile Tyr Phe Cys Ala Glu Ser Ile Arg
435 440 445
Gly Phe Gly Asn Val Leu His Cys Gly Ser Gly Thr Gln Val Ile Val
450 455 460
Leu Pro His Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp
465 470 475 480
Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser
485 490 495
Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp
500 505 510
Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala
515 520 525
Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn
530 535 540
Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser
545 550 555 560
Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu
565 570 575
Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys
580 585 590
Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600 605
<210> 227
<211> 606
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 7/ TCR 54 - (E7)11 - полноразмерная последовательность, нативный
<400> 227
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ile Thr Asp Arg Thr Asn Tyr Gly Tyr Thr Phe Gly Ser Gly Thr Arg
115 120 125
Leu Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Phe Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu Lys
305 310 315 320
Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Lys Lys His Leu
325 330 335
Thr Thr Phe Leu Val Ile Leu Trp Leu Tyr Phe Tyr Arg Gly Asn Gly
340 345 350
Lys Asn Gln Val Glu Gln Ser Pro Gln Ser Leu Ile Ile Leu Glu Gly
355 360 365
Lys Asn Cys Thr Leu Gln Cys Asn Tyr Thr Val Ser Pro Phe Ser Asn
370 375 380
Leu Arg Trp Tyr Lys Gln Asp Thr Gly Arg Gly Pro Val Ser Leu Thr
385 390 395 400
Ile Met Thr Phe Ser Glu Asn Thr Lys Ser Asn Gly Arg Tyr Thr Ala
405 410 415
Thr Leu Asp Ala Asp Thr Lys Gln Ser Ser Leu His Ile Thr Ala Ser
420 425 430
Gln Leu Ser Asp Ser Ala Ser Tyr Ile Cys Val Val Ser Arg Asp Asn
435 440 445
Tyr Gly Gln Asn Phe Val Phe Gly Pro Gly Thr Arg Leu Ser Val Leu
450 455 460
Pro Tyr Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser
465 470 475 480
Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln
485 490 495
Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys
500 505 510
Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val
515 520 525
Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn
530 535 540
Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys
545 550 555 560
Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn
565 570 575
Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val
580 585 590
Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600 605
<210> 228
<211> 606
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 8 полноразмерная последовательность, нативный
<400> 228
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Ser Leu Trp Gly Ala Ser Thr Asp Thr Gln Tyr Phe Gly Pro Gly Thr
115 120 125
Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser
305 310 315 320
Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Glu
325 330 335
Thr Leu Leu Gly Leu Leu Ile Leu Trp Leu Gln Leu Gln Trp Val Ser
340 345 350
Ser Lys Gln Glu Val Thr Gln Ile Pro Ala Ala Leu Ser Val Pro Glu
355 360 365
Gly Glu Asn Leu Val Leu Asn Cys Ser Phe Thr Asp Ser Ala Ile Tyr
370 375 380
Asn Leu Gln Trp Phe Arg Gln Asp Pro Gly Lys Gly Leu Thr Ser Leu
385 390 395 400
Leu Leu Ile Gln Ser Ser Gln Arg Glu Gln Thr Ser Gly Arg Leu Asn
405 410 415
Ala Ser Leu Asp Lys Ser Ser Gly Arg Ser Thr Leu Tyr Ile Ala Ala
420 425 430
Ser Gln Pro Gly Asp Ser Ala Thr Tyr Leu Cys Ala Val Arg Pro Leu
435 440 445
Gly Asn Thr Pro Leu Val Phe Gly Lys Gly Thr Arg Leu Ser Val Ile
450 455 460
Ala Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser
465 470 475 480
Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln
485 490 495
Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys
500 505 510
Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val
515 520 525
Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn
530 535 540
Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys
545 550 555 560
Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn
565 570 575
Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val
580 585 590
Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600 605
<210> 229
<211> 611
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 9 - полноразмерная последовательность, нативный
<400> 229
Met Ser Leu Gly Leu Leu Cys Cys Gly Ala Phe Ser Leu Leu Trp Ala
1 5 10 15
Gly Pro Val Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Arg Val Leu
20 25 30
Lys Thr Gly Gln Ser Met Thr Leu Leu Cys Ala Gln Asp Met Asn His
35 40 45
Glu Tyr Met Tyr Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu
50 55 60
Ile His Tyr Ser Val Gly Glu Gly Thr Thr Ala Lys Gly Glu Val Pro
65 70 75 80
Asp Gly Tyr Asn Val Ser Arg Leu Lys Lys Gln Asn Phe Leu Leu Gly
85 90 95
Leu Glu Ser Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser
100 105 110
Ser Tyr Phe Gly Thr Ala Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser Leu
305 310 315 320
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Ser Leu
325 330 335
Ser Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu Gly Pro Gly
340 345 350
Ile Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu
355 360 365
Lys Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Pro Ser
370 375 380
Tyr Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe
385 390 395 400
Leu Ile Tyr Gln Gly Ser Tyr Asp Gln Gln Asn Ala Thr Glu Gly Arg
405 410 415
Tyr Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile
420 425 430
Ser Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Arg
435 440 445
Thr Ala Gly Gly Thr Ser Tyr Gly Lys Leu Thr Phe Gly Gln Gly Thr
450 455 460
Ile Leu Thr Val His Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr
465 470 475 480
Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr
485 490 495
Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val
500 505 510
Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys
515 520 525
Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala
530 535 540
Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser
545 550 555 560
Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr
565 570 575
Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile
580 585 590
Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu
595 600 605
Trp Ser Ser
610
<210> 230
<211> 612
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 10 полноразмерная последовательность, нативный
<400> 230
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Ser Leu Ser Leu Thr Gly Asn Tyr Gly Tyr Thr Phe Gly Ser Gly Thr
115 120 125
Arg Leu Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu
305 310 315 320
Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Met Ile Ser
325 330 335
Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu Ser Trp Val Trp
340 345 350
Ser Gln Arg Lys Glu Val Glu Gln Asp Pro Gly Pro Phe Asn Val Pro
355 360 365
Glu Gly Ala Thr Val Ala Phe Asn Cys Thr Tyr Ser Asn Ser Ala Ser
370 375 380
Gln Ser Phe Phe Trp Tyr Arg Gln Asp Cys Arg Lys Glu Pro Lys Leu
385 390 395 400
Leu Met Ser Val Tyr Ser Ser Gly Asn Glu Asp Gly Arg Phe Thr Ala
405 410 415
Gln Leu Asn Arg Ala Ser Gln Tyr Ile Ser Leu Leu Ile Arg Asp Ser
420 425 430
Lys Leu Ser Asp Ser Ala Thr Tyr Leu Cys Val Val Asn Phe Pro Ser
435 440 445
Arg Gly Ala Gly Gly Thr Ser Tyr Gly Lys Leu Thr Phe Gly Gln Gly
450 455 460
Thr Ile Leu Thr Val His Pro Asn Ile Gln Lys Pro Asp Pro Ala Val
465 470 475 480
Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe
485 490 495
Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp
500 505 510
Val Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe
515 520 525
Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys
530 535 540
Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Ala Asp Thr Phe Phe Pro
545 550 555 560
Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu
565 570 575
Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg
580 585 590
Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg
595 600 605
Leu Trp Ser Ser
610
<210> 231
<211> 604
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 11 полноразмерная последовательность, нативный
<400> 231
Met Leu Ser Leu Leu Leu Leu Leu Leu Gly Leu Gly Ser Val Phe Ser
1 5 10 15
Ala Val Ile Ser Gln Lys Pro Ser Arg Asp Ile Cys Gln Arg Gly Thr
20 25 30
Ser Leu Thr Ile Gln Cys Gln Val Asp Ser Gln Val Thr Met Met Phe
35 40 45
Trp Tyr Arg Gln Gln Pro Gly Gln Ser Leu Thr Leu Ile Ala Thr Ala
50 55 60
Asn Gln Gly Ser Glu Ala Thr Tyr Glu Ser Gly Phe Val Ile Asp Lys
65 70 75 80
Phe Pro Ile Ser Arg Pro Asn Leu Thr Phe Ser Thr Leu Thr Val Ser
85 90 95
Asn Met Ser Pro Glu Asp Ser Ser Ile Tyr Leu Cys Ser Val Val Pro
100 105 110
Trp Thr Arg Gly Gly Ser Thr Asp Thr Gln Tyr Phe Gly Pro Gly Thr
115 120 125
Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser
305 310 315 320
Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Lys
325 330 335
Leu Val Thr Ser Ile Thr Val Leu Leu Ser Leu Gly Ile Met Gly Asp
340 345 350
Ala Lys Thr Thr Gln Pro Asn Ser Met Glu Ser Asn Glu Glu Glu Pro
355 360 365
Val His Leu Pro Cys Asn His Ser Thr Ile Ser Gly Thr Asp Tyr Ile
370 375 380
His Trp Tyr Arg Gln Leu Pro Ser Gln Gly Pro Glu Tyr Val Ile His
385 390 395 400
Gly Leu Thr Ser Asn Val Asn Asn Arg Met Ala Ser Leu Ala Ile Ala
405 410 415
Glu Asp Arg Lys Ser Ser Thr Leu Ile Leu His Arg Ala Thr Leu Arg
420 425 430
Asp Ala Ala Val Tyr Tyr Cys Ile Leu Ser Ala His Ser Asn Ser Gly
435 440 445
Tyr Ala Leu Asn Phe Gly Lys Gly Thr Ser Leu Leu Val Thr Pro His
450 455 460
Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser
465 470 475 480
Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn
485 490 495
Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr Val
500 505 510
Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp
515 520 525
Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile
530 535 540
Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val
545 550 555 560
Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln
565 570 575
Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly
580 585 590
Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600
<210> 232
<211> 9
<212> Белок
<213> Вирус папилломы человека
<220>
<223> E6(18-26) пептид
<400> 232
Lys Leu Pro Gln Leu Cys Thr Glu Leu
1 5
<210> 233
<211> 10
<212> Белок
<213> Вирус папилломы человека
<220>
<223> E6(29-38) пептид
<400> 233
Thr Ile His Asp Ile Ile Leu Glu Cys Val
1 5 10
<210> 234
<211> 9
<212> Белок
<213> Вирус папилломы человека
<220>
<223> E6(52-60) пептид
<400> 234
Phe Ala Phe Arg Asp Leu Cys Ile Val
1 5
<210> 235
<211> 8
<212> Белок
<213> Вирус папилломы человека
<220>
<223> E7(86-93) пептид
<400> 235
Thr Leu Gly Ile Val Cys Pro Ile
1 5
<210> 236
<211> 9
<212> Белок
<213> Вирус папилломы человека
<220>
<223> E7(11-19) пептид
<400> 236
Tyr Met Leu Asp Leu Gln Pro Glu Thr
1 5
<210> 237
<211> 9
<212> Белок
<213> Вирус папилломы человека
<220>
<223> E7(85-93) пептид
<400> 237
Gly Thr Leu Gly Ile Val Cys Pro Ile
1 5
<210> 238
<211> 9
<212> Белок
<213> Вирус папилломы человека
<220>
<223> E7(82-90) пептид
<400> 238
Leu Leu Met Gly Thr Leu Gly Ile Val
1 5
<210> 239
<211> 9
<212> Белок
<213> Вирус папилломы человека
<220>
<223> E7(7-15) пептид
<400> 239
Thr Leu His Glu Tyr Met Leu Asp Leu
1 5
<210> 240
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR альфа E6(29-38) CDR1 консенсус
<220>
<221> ВАРИАНТ
<222> (1)...(1)
<223> X1 = T, D, S или N
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 = I или S
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 = S, D, N, Y или A
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 = G, Q, P или отсутствует
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 = T, S, F или I
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 = D, Y, P или Q
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 = Y, G, N, A, S или Q
<400> 240
Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5
<210> 241
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR альфа E7(11-19) CDR1 консенсус
<220>
<221> ВАРИАНТ
<222> (1)...(1)
<223> X1 = D или V
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 = S или P
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 = S или F
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 = T или S
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 = Y или N
<400> 241
Xaa Ser Xaa Xaa Xaa Xaa
1 5
<210> 242
<211> 273
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 12 / TCR 55 - (E7)11 - альфа
цистеин-модифицированный
<400> 242
Met Lys Thr Phe Ala Gly Phe Ser Phe Leu Phe Leu Trp Leu Gln Leu
1 5 10 15
Asp Cys Met Ser Arg Gly Glu Asp Val Glu Gln Ser Leu Phe Leu Ser
20 25 30
Val Arg Glu Gly Asp Ser Ser Val Ile Asn Cys Thr Tyr Thr Asp Ser
35 40 45
Ser Ser Thr Tyr Leu Tyr Trp Tyr Lys Gln Glu Pro Gly Ala Gly Leu
50 55 60
Gln Leu Leu Thr Tyr Ile Phe Ser Asn Met Asp Met Lys Gln Asp Gln
65 70 75 80
Arg Leu Thr Val Leu Leu Asn Lys Lys Asp Lys His Leu Ser Leu Arg
85 90 95
Ile Ala Asp Thr Gln Thr Gly Asp Ser Ala Ile Tyr Phe Cys Ala Val
100 105 110
Pro Ser Gly Ala Thr Asn Lys Leu Ile Phe Gly Thr Gly Thr Leu Leu
115 120 125
Ala Val Gln Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu
130 135 140
Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe
145 150 155 160
Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile
165 170 175
Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn
180 185 190
Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala
195 200 205
Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu
210 215 220
Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr
225 230 235 240
Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu
245 250 255
Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser
260 265 270
Ser
<210> 243
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR альфа, общий консенсус CDR1
<220>
<221> ВАРИАНТ
<222> (1)...(1)
<223> X1 = T, D, N, S или V
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 = I или S
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 = S, D, A, P, N или Y
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 = G, Q, P или отсутствует
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 = T, S, I или F
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 = D, Y, ,Q, T, P или S
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 = Y, G, N, A, S или Q
<400> 243
Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5
<210> 244
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR альфа E6(29-38) CDR2 консенсус
<220>
<221> ВАРИАНТ
<222> (1)...(1)
<223> X1 = G, Q, I, M, Y или V
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 = L, S, Q, T или Y
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 = T, G, L или S
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 = Y, S, N, A или отсутствует
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 = отсутствует, A или D
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 = отсутствует, E, Q, T или S
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 = S, Q, R, L или G
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X8 = N, V или E
<400> 244
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5
<210> 245
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR альфа E7(11-19) CDR2 консенсус
<220>
<221> ВАРИАНТ
<222> (1)...(1)
<223> X1 = I или M
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 = F или T
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 = S или F
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 = N или S
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 = M или E
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 = D или N
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 = M или T
<400> 245
Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5
<210> 246
<211> 311
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 6, TCR 12, TCR 55 - (E7)11 -бета
нативный
<400> 246
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ser Thr Thr Arg Ser Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 247
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR альфа, общий консенсус CDR2
<220>
<221> ВАРИАНТ
<222> (1)...(1)
<223> X1 = G, Q, I, V, Y или M
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 = L, S, Q, Y, F или T
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 = T, G, S, L или F
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 = Y, S, N, A или отсутствует
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 = отсутствует, A или D
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 = отсутствует, E, Q, S, M или T
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 = S, Q, R, G, D, L или N
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X8 = N, E, M, T или V
<400> 247
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5
<210> 248
<211> 18
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR альфа E6(29-38) CDR3 консенсус
<220>
<221> ВАРИАНТ
<222> (1)...(1)
<223> X1 = A, I или V
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 = M, L, S или V
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 = R, L, Q или N
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 = E, V, T, P, G или F
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 = G, I, L, A, отсутствует или P
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 = R, T, G, отсутствует или S
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 = G, R или отсутствует
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X8 = T, G или отсутствует
<220>
<221> ВАРИАНТ
<222> (9)...(9)
<223> X9 = отсутствует или A
<220>
<221> ВАРИАНТ
<222> (10)...(10)
<223> X10 = отсутствует или G
<220>
<221> ВАРИАНТ
<222> (11)...(11)
<223> X11 = отсутствует или G
<220>
<221> ВАРИАНТ
<222> (12)...(12)
<223> X12 = отсутствует или T
<220>
<221> ВАРИАНТ
<222> (13)...(13)
<223> X13 = отсутствует или S
<220>
<221> ВАРИАНТ
<222> (14)...(14)
<223> X14 = G, Y, отсутствует или N
<220>
<221> ВАРИАНТ
<222> (15)...(15)
<223> X15 = F, G, N или T
<220>
<221> ВАРИАНТ
<222> (16)...(16)
<223> X16 = K или N, P
<220>
<221> ВАРИАНТ
<222> (17)...(18)
<223> X17 = T или L
X18 = I, V, F или T
<400> 248
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5 10 15
Xaa Xaa
<210> 249
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR альфа E7(11-19) CDR3 консенсус
<220>
<221> ВАРИАНТ
<222> (1)...(1)
<223> X1 = A или V
<220>
<221> ВАРИАНТ
<222> (2)...(3)
<223> X2 = E или V
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 = S или P
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 = I, S или R
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 = R, G или D
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 = G, A или N
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 = F, отсутствует или Y
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X8 = G или T
<220>
<221> ВАРИАНТ
<222> (9)...(9)
<223> X9 = N, T или Q
<220>
<221> ВАРИАНТ
<222> (10)...(10)
<223> X10 = V, K или N
<220>
<221> ВАРИАНТ
<222> (11)...(11)
<223> X11 = L или F
<220>
<221> ВАРИАНТ
<222> (12)...(12)
<223> X12 = H, I или V
<400> 249
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5 10
<210> 250
<211> 311
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 6, TCR 12, TCR 55 - (E7)11 - бета
цистеин-модифицированный
<400> 250
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ser Thr Thr Arg Ser Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 251
<211> 18
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR альфа, общий консенсус CDR3
<220>
<221> ВАРИАНТ
<222> (1)...(1)
<223> X1 = A, I или V
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 = M, L, V, E или S
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 = R, L, N, Q, P или S
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 = E, V, P, T, F, I, R, G, S или A
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 = G, I, L, A, P, R, D, отсутствует или H
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 = R, T, G, S, N, отсутствует или A
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 = G, R, N или отсутствует
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X8 = T, G или отсутствует
<220>
<221> ВАРИАНТ
<222> (9)...(9)
<223> X9 = отсутствует или A
<220>
<221> ВАРИАНТ
<222> (10)...(10)
<223> X10 = отсутствует или G
<220>
<221> ВАРИАНТ
<222> (11)...(11)
<223> X11 = отсутствует или G
<220>
<221> ВАРИАНТ
<222> (12)...(12)
<223> X12 = отсутствует или T
<220>
<221> ВАРИАНТ
<222> (13)...(13)
<223> X13 = F, Y, S или отсутствует
<220>
<221> ВАРИАНТ
<222> (14)...(14)
<223> X14 = G, Y, отсутствует или N
<220>
<221> ВАРИАНТ
<222> (15)...(15)
<223> X15 = F, G, T, N, Q или Y
<220>
<221> ВАРИАНТ
<222> (16)...(16)
<223> X16 = K, P, V, N или A
<220>
<221> ВАРИАНТ
<222> (17)...(18)
<223> X17 = T, L или F
X18 = I, V, T, H, F или N
<400> 251
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5 10 15
Xaa Xaa
<210> 252
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR бета E6(29-38) CDR1 консенсус
<220>
<221> ВАРИАНТ
<222> (1)...(1)
<223> X1 = S или M
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 = G, E, D или N
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 = V, N или E
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 = S, R, N или Y
<400> 252
Xaa Xaa His Xaa Xaa
1 5
<210> 253
<211> 270
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 13 - альфа нативный
<400> 253
Met Leu Leu Leu Leu Val Pro Val Leu Glu Val Ile Phe Thr Leu Gly
1 5 10 15
Gly Thr Arg Ala Gln Ser Val Thr Gln Leu Asp Ser His Val Ser Val
20 25 30
Ser Glu Gly Thr Pro Val Leu Leu Arg Cys Asn Tyr Ser Ser Ser Tyr
35 40 45
Ser Pro Ser Leu Phe Trp Tyr Val Gln His Pro Asn Lys Gly Leu Gln
50 55 60
Leu Leu Leu Lys Tyr Thr Ser Ala Ala Thr Leu Val Lys Gly Ile Asn
65 70 75 80
Gly Phe Glu Ala Glu Phe Lys Lys Ser Glu Thr Ser Phe His Leu Thr
85 90 95
Lys Pro Ser Ala His Met Ser Asp Ala Ala Glu Tyr Phe Cys Val Val
100 105 110
Arg Gly Gly Lys Leu Ile Phe Gly Gln Gly Thr Glu Leu Ser Val Lys
115 120 125
Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser
130 135 140
Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln
145 150 155 160
Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys
165 170 175
Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val
180 185 190
Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn
195 200 205
Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys
210 215 220
Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn
225 230 235 240
Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val
245 250 255
Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
260 265 270
<210> 254
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR бета общий консенсус CDR1
<220>
<221> ВАРИАНТ
<222> (1)...(1)
<223> X1 = S или M
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 = G, E, D, N или Q
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 = H или V
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 = V, N, E, L или T
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 = S, R, N, Y или M
<400> 254
Xaa Xaa Xaa Xaa Xaa
1 5
<210> 255
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR бета E6(29-38) CDR2 консенсус
<220>
<221> ВАРИАНТ
<222> (1)...(1)
<223> X1 = F или S
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 = Q, Y или V
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 = N, D или G
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 = E или V
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 = A, K или G
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 = Q, M или T
<400> 255
Xaa Xaa Xaa Xaa Xaa Xaa
1 5
<210> 256
<211> 270
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 13 - альфа, цистеин-модифицированный
<400> 256
Met Leu Leu Leu Leu Val Pro Val Leu Glu Val Ile Phe Thr Leu Gly
1 5 10 15
Gly Thr Arg Ala Gln Ser Val Thr Gln Leu Asp Ser His Val Ser Val
20 25 30
Ser Glu Gly Thr Pro Val Leu Leu Arg Cys Asn Tyr Ser Ser Ser Tyr
35 40 45
Ser Pro Ser Leu Phe Trp Tyr Val Gln His Pro Asn Lys Gly Leu Gln
50 55 60
Leu Leu Leu Lys Tyr Thr Ser Ala Ala Thr Leu Val Lys Gly Ile Asn
65 70 75 80
Gly Phe Glu Ala Glu Phe Lys Lys Ser Glu Thr Ser Phe His Leu Thr
85 90 95
Lys Pro Ser Ala His Met Ser Asp Ala Ala Glu Tyr Phe Cys Val Val
100 105 110
Arg Gly Gly Lys Leu Ile Phe Gly Gln Gly Thr Glu Leu Ser Val Lys
115 120 125
Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser
130 135 140
Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln
145 150 155 160
Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys
165 170 175
Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val
180 185 190
Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn
195 200 205
Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys
210 215 220
Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn
225 230 235 240
Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val
245 250 255
Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
260 265 270
<210> 257
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR бета, общий консенсус CDR2
<220>
<221> ВАРИАНТ
<222> (1)...(1)
<223> X1 = F, S или A
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 = Q, Y, V или N
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 = N, D, G или Q
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 = E, V, N или S
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 = A, K, G или E
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 = Q, M, T, I или A
<400> 257
Xaa Xaa Xaa Gly Xaa Xaa Xaa
1 5
<210> 258
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR бета E6(29-38) CDR3 консенсус
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 = S или T
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 = H, P, L, F или Y
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 = L, G, W, F, T или S
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 = A, G или L
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 = G, E, A, T, Q или отсутствует
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X8 = F, G, T, R или S
<220>
<221> ВАРИАНТ
<222> (9)...(9)
<223> X9 = T, N, H, R, E или A
<220>
<221> ВАРИАНТ
<222> (10)...(10)
<223> X10 = G, T, Q, D, R или Y
<220>
<221> ВАРИАНТ
<222> (11)...(11)
<223> X11 = E, P, T или G
<220>
<221> ВАРИАНТ
<222> (12)...(12)
<223> X12 = L, A, Q или Y
<220>
<221> ВАРИАНТ
<222> (13)...(13)
<223> X13 = F, H, Y или T
<400> 258
Ala Ser Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5 10
<210> 259
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR бета E7(11-19) CDR3 консенсус
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 = S или I
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 = T или D
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 = S или T
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 = S или N
<220>
<221> ВАРИАНТ
<222> (9)...(9)
<223> X9 = E или G
<220>
<221> ВАРИАНТ
<222> (10)...(10)
<223> X10 = Q или Y
<220>
<221> ВАРИАНТ
<222> (11)...(11)
<223> X11 = Y или T
<400> 259
Ala Xaa Thr Xaa Arg Xaa Xaa Tyr Xaa Xaa Xaa
1 5 10
<210> 260
<211> 312
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 13 - бета нативный
<400> 260
Met Gly Thr Arg Leu Leu Cys Trp Val Val Leu Gly Phe Leu Gly Thr
1 5 10 15
Asp His Thr Gly Ala Gly Val Ser Gln Ser Pro Arg Tyr Lys Val Ala
20 25 30
Lys Arg Gly Gln Asp Val Ala Leu Arg Cys Asp Pro Ile Ser Gly His
35 40 45
Val Ser Leu Phe Trp Tyr Gln Gln Ala Leu Gly Gln Gly Pro Glu Phe
50 55 60
Leu Thr Tyr Phe Gln Asn Glu Ala Gln Leu Asp Lys Ser Gly Leu Pro
65 70 75 80
Ser Asp Arg Phe Phe Ala Glu Arg Pro Glu Gly Ser Val Ser Thr Leu
85 90 95
Lys Ile Gln Arg Thr Gln Gln Glu Asp Ser Ala Val Tyr Leu Cys Ala
100 105 110
Ser Ser Pro Thr Gly Thr Glu Arg Glu Leu Phe Phe Gly Glu Gly Ser
115 120 125
Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 261
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR бета, общий консенсус CDR3
<220>
<221> ВАРИАНТ
<222> (1)...(1)
<223> X1 = A или S
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 = S, I или V
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 = S, T или V
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 = H, P, L, Y, T, D или F
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 = L, G, W, F, S, T или R
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 = A, G, L, S или T
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 = G, E, A, T, R, Q или отсутствует
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X8 = отсутствует или G
<220>
<221> ВАРИАНТ
<222> (9)...(9)
<223> X9 = отсутствует или G
<220>
<221> ВАРИАНТ
<222> (10)...(10)
<223> X10 = отсутствует, F, G, T, S или R
<220>
<221> ВАРИАНТ
<222> (11)...(11)
<223> X11 = T, N, H, A, S, R или E
<220>
<221> ВАРИАНТ
<222> (12)...(12)
<223> X12 = G, T, Q, D, Y или R
<220>
<221> ВАРИАНТ
<222> (13)...(13)
<223> X13 = E, P, T или G
<220>
<221> ВАРИАНТ
<222> (14)...(14)
<223> X14 = L, A, Q или Y
<220>
<221> ВАРИАНТ
<222> (15)...(15)
<223> X15 = F, H, Y или T
<400> 261
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5 10 15
<210> 262
<211> 137
<212> Белок
<213> Mus musculus
<220>
<223> Мышиная константная область альфа
<400> 262
Asp Ile Gln Asn Pro Glu Pro Ala Val Tyr Gln Leu Lys Asp Pro Arg
1 5 10 15
Ser Gln Asp Ser Thr Leu Cys Leu Phe Thr Asp Phe Asp Ser Gln Ile
20 25 30
Asn Val Pro Lys Thr Met Glu Ser Gly Thr Phe Ile Thr Asp Lys Thr
35 40 45
Val Leu Asp Met Lys Ala Met Asp Ser Lys Ser Asn Gly Ala Ile Ala
50 55 60
Trp Ser Asn Gln Thr Ser Phe Thr Cys Gln Asp Ile Phe Lys Glu Thr
65 70 75 80
Asn Ala Thr Tyr Pro Ser Ser Asp Val Pro Cys Asp Ala Thr Leu Thr
85 90 95
Glu Lys Ser Phe Glu Thr Asp Met Asn Leu Asn Phe Gln Asn Leu Ser
100 105 110
Val Met Gly Leu Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu
115 120 125
Leu Met Thr Leu Arg Leu Trp Ser Ser
130 135
<210> 263
<211> 173
<212> Белок
<213> Mus musculus
<220>
<223> Мышиная константная область бета
<400> 263
Glu Asp Leu Arg Asn Val Thr Pro Pro Lys Val Ser Leu Phe Glu Pro
1 5 10 15
Ser Lys Ala Glu Ile Ala Asn Lys Gln Lys Ala Thr Leu Val Cys Leu
20 25 30
Ala Arg Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp Trp Val Asn
35 40 45
Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Ala Tyr Lys
50 55 60
Glu Ser Asn Tyr Ser Tyr Cys Leu Ser Ser Arg Leu Arg Val Ser Ala
65 70 75 80
Thr Phe Trp His Asn Pro Arg Asn His Phe Arg Cys Gln Val Gln Phe
85 90 95
His Gly Leu Ser Glu Glu Asp Lys Trp Pro Glu Gly Ser Pro Lys Pro
100 105 110
Val Thr Gln Asn Ile Ser Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly
115 120 125
Ile Thr Ser Ala Ser Tyr Gln Gln Gly Val Leu Ser Ala Thr Ile Leu
130 135 140
Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr Ala Val Leu Val Ser
145 150 155 160
Thr Leu Val Val Met Ala Met Val Lys Arg Lys Asn Ser
165 170
<210> 264
<211> 158
<212> Белок
<213> Вирус папилломы человека
<220>
<223> HPV 16 E6
<400> 264
Met His Gln Lys Arg Thr Ala Met Phe Gln Asp Pro Gln Glu Arg Pro
1 5 10 15
Arg Lys Leu Pro Gln Leu Cys Thr Glu Leu Gln Thr Thr Ile His Asp
20 25 30
Ile Ile Leu Glu Cys Val Tyr Cys Lys Gln Gln Leu Leu Arg Arg Glu
35 40 45
Val Tyr Asp Phe Ala Phe Arg Asp Leu Cys Ile Val Tyr Arg Asp Gly
50 55 60
Asn Pro Tyr Ala Val Cys Asp Lys Cys Leu Lys Phe Tyr Ser Lys Ile
65 70 75 80
Ser Glu Tyr Arg His Tyr Cys Tyr Ser Leu Tyr Gly Thr Thr Leu Glu
85 90 95
Gln Gln Tyr Asn Lys Pro Leu Cys Asp Leu Leu Ile Arg Cys Ile Asn
100 105 110
Cys Gln Lys Pro Leu Cys Pro Glu Glu Lys Gln Arg His Leu Asp Lys
115 120 125
Lys Gln Arg Phe His Asn Ile Arg Gly Arg Trp Thr Gly Arg Cys Met
130 135 140
Ser Cys Cys Arg Ser Ser Arg Thr Arg Arg Glu Thr Gln Leu
145 150 155
<210> 265
<211> 98
<212> Белок
<213> Вирус папилломы человека
<220>
<223> HPV 16 E7
<400> 265
Met His Gly Asp Thr Pro Thr Leu His Glu Tyr Met Leu Asp Leu Gln
1 5 10 15
Pro Glu Thr Thr Asp Leu Tyr Cys Tyr Glu Gln Leu Asn Asp Ser Ser
20 25 30
Glu Glu Glu Asp Glu Ile Asp Gly Pro Ala Gly Gln Ala Glu Pro Asp
35 40 45
Arg Ala His Tyr Asn Ile Val Thr Phe Cys Cys Lys Cys Asp Ser Thr
50 55 60
Leu Arg Leu Cys Val Gln Ser Thr His Val Asp Ile Arg Thr Leu Glu
65 70 75 80
Asp Leu Leu Met Gly Thr Leu Gly Ile Val Cys Pro Ile Cys Ser Gln
85 90 95
Lys Pro
<210> 266
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> Линкер
<220>
<221> ПОВТОР
<222> (5)...(9)
<223> Повторяется 5 или 6 раз
<400> 266
Pro Gly Gly Gly Ser Gly Gly Gly Gly Pro
1 5 10
<210> 267
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> Линкер
<400> 267
Gly Ser Ala Asp Asp Ala Lys Lys Asp Ala Ala Lys Lys Asp Gly Lys
1 5 10 15
Ser
<210> 268
<211> 12
<212> Белок
<213> Homo sapiens
<220>
<223> Спейсер (IgG4шарнир)
<400> 268
Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro
1 5 10
<210> 269
<211> 36
<212> ДНК
<213> Homo sapiens
<220>
<223> Спейсер (IgG4шарнир)
<400> 269
gaatctaagt acggaccgcc ctgcccccct tgccct 36
<210> 270
<211> 119
<212> Белок
<213> Homo sapiens
<220>
<223> шарнир-CH3 Спейсер
<400> 270
Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro Gly Gln Pro Arg
1 5 10 15
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys
20 25 30
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
35 40 45
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
50 55 60
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
65 70 75 80
Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser
85 90 95
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
100 105 110
Leu Ser Leu Ser Leu Gly Lys
115
<210> 271
<211> 229
<212> Белок
<213> Homo sapiens
<220>
<223> шарнир-CH2-CH3 Спейсер
<400> 271
Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro Ala Pro Glu Phe
1 5 10 15
Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
20 25 30
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
35 40 45
Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val
50 55 60
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser
65 70 75 80
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
85 90 95
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser
100 105 110
Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
115 120 125
Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln
130 135 140
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
145 150 155 160
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
165 170 175
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu
180 185 190
Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser
195 200 205
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
210 215 220
Leu Ser Leu Gly Lys
225
<210> 272
<211> 282
<212> Белок
<213> Homo sapiens
<220>
<223> IgD-шарнир-Fc
<400> 272
Arg Trp Pro Glu Ser Pro Lys Ala Gln Ala Ser Ser Val Pro Thr Ala
1 5 10 15
Gln Pro Gln Ala Glu Gly Ser Leu Ala Lys Ala Thr Thr Ala Pro Ala
20 25 30
Thr Thr Arg Asn Thr Gly Arg Gly Gly Glu Glu Lys Lys Lys Glu Lys
35 40 45
Glu Lys Glu Glu Gln Glu Glu Arg Glu Thr Lys Thr Pro Glu Cys Pro
50 55 60
Ser His Thr Gln Pro Leu Gly Val Tyr Leu Leu Thr Pro Ala Val Gln
65 70 75 80
Asp Leu Trp Leu Arg Asp Lys Ala Thr Phe Thr Cys Phe Val Val Gly
85 90 95
Ser Asp Leu Lys Asp Ala His Leu Thr Trp Glu Val Ala Gly Lys Val
100 105 110
Pro Thr Gly Gly Val Glu Glu Gly Leu Leu Glu Arg His Ser Asn Gly
115 120 125
Ser Gln Ser Gln His Ser Arg Leu Thr Leu Pro Arg Ser Leu Trp Asn
130 135 140
Ala Gly Thr Ser Val Thr Cys Thr Leu Asn His Pro Ser Leu Pro Pro
145 150 155 160
Gln Arg Leu Met Ala Leu Arg Glu Pro Ala Ala Gln Ala Pro Val Lys
165 170 175
Leu Ser Leu Asn Leu Leu Ala Ser Ser Asp Pro Pro Glu Ala Ala Ser
180 185 190
Trp Leu Leu Cys Glu Val Ser Gly Phe Ser Pro Pro Asn Ile Leu Leu
195 200 205
Met Trp Leu Glu Asp Gln Arg Glu Val Asn Thr Ser Gly Phe Ala Pro
210 215 220
Ala Arg Pro Pro Pro Gln Pro Gly Ser Thr Thr Phe Trp Ala Trp Ser
225 230 235 240
Val Leu Arg Val Pro Ala Pro Pro Ser Pro Gln Pro Ala Thr Tyr Thr
245 250 255
Cys Val Val Ser His Glu Asp Ser Arg Thr Leu Leu Asn Ala Ser Arg
260 265 270
Ser Leu Glu Val Ser Tyr Val Thr Asp His
275 280
<210> 273
<211> 357
<212> Белок
<213> Искусственная последовательность
<220>
<223> tEGFR
<400> 273
Met Leu Leu Leu Val Thr Ser Leu Leu Leu Cys Glu Leu Pro His Pro
1 5 10 15
Ala Phe Leu Leu Ile Pro Arg Lys Val Cys Asn Gly Ile Gly Ile Gly
20 25 30
Glu Phe Lys Asp Ser Leu Ser Ile Asn Ala Thr Asn Ile Lys His Phe
35 40 45
Lys Asn Cys Thr Ser Ile Ser Gly Asp Leu His Ile Leu Pro Val Ala
50 55 60
Phe Arg Gly Asp Ser Phe Thr His Thr Pro Pro Leu Asp Pro Gln Glu
65 70 75 80
Leu Asp Ile Leu Lys Thr Val Lys Glu Ile Thr Gly Phe Leu Leu Ile
85 90 95
Gln Ala Trp Pro Glu Asn Arg Thr Asp Leu His Ala Phe Glu Asn Leu
100 105 110
Glu Ile Ile Arg Gly Arg Thr Lys Gln His Gly Gln Phe Ser Leu Ala
115 120 125
Val Val Ser Leu Asn Ile Thr Ser Leu Gly Leu Arg Ser Leu Lys Glu
130 135 140
Ile Ser Asp Gly Asp Val Ile Ile Ser Gly Asn Lys Asn Leu Cys Tyr
145 150 155 160
Ala Asn Thr Ile Asn Trp Lys Lys Leu Phe Gly Thr Ser Gly Gln Lys
165 170 175
Thr Lys Ile Ile Ser Asn Arg Gly Glu Asn Ser Cys Lys Ala Thr Gly
180 185 190
Gln Val Cys His Ala Leu Cys Ser Pro Glu Gly Cys Trp Gly Pro Glu
195 200 205
Pro Arg Asp Cys Val Ser Cys Arg Asn Val Ser Arg Gly Arg Glu Cys
210 215 220
Val Asp Lys Cys Asn Leu Leu Glu Gly Glu Pro Arg Glu Phe Val Glu
225 230 235 240
Asn Ser Glu Cys Ile Gln Cys His Pro Glu Cys Leu Pro Gln Ala Met
245 250 255
Asn Ile Thr Cys Thr Gly Arg Gly Pro Asp Asn Cys Ile Gln Cys Ala
260 265 270
His Tyr Ile Asp Gly Pro His Cys Val Lys Thr Cys Pro Ala Gly Val
275 280 285
Met Gly Glu Asn Asn Thr Leu Val Trp Lys Tyr Ala Asp Ala Gly His
290 295 300
Val Cys His Leu Cys His Pro Asn Cys Thr Tyr Gly Cys Thr Gly Pro
305 310 315 320
Gly Leu Glu Gly Cys Pro Thr Asn Gly Pro Lys Ile Pro Ser Ile Ala
325 330 335
Thr Gly Met Val Gly Ala Leu Leu Leu Leu Leu Val Val Ala Leu Gly
340 345 350
Ile Gly Leu Phe Met
355
<210> 274
<211> 24
<212> Белок
<213> Искусственная последовательность
<220>
<223> T2A
<400> 274
Leu Glu Gly Gly Gly Glu Gly Arg Gly Ser Leu Leu Thr Cys Gly Asp
1 5 10 15
Val Glu Glu Asn Pro Gly Pro Arg
20
<210> 275
<211> 27
<212> Белок
<213> Homo sapiens
<220>
<223> CD28 (аминокислоты 153-179 регистр. No. P10747)
<400> 275
Phe Trp Val Leu Val Val Val Gly Gly Val Leu Ala Cys Tyr Ser Leu
1 5 10 15
Leu Val Thr Val Ala Phe Ile Ile Phe Trp Val
20 25
<210> 276
<211> 66
<212> Белок
<213> Homo sapiens
<220>
<223> CD28 (аминокислоты 114-179 регистр. No. P10747)
<400> 276
Ile Glu Val Met Tyr Pro Pro Pro Tyr Leu Asp Asn Glu Lys Ser Asn
1 5 10 15
Gly Thr Ile Ile His Val Lys Gly Lys His Leu Cys Pro Ser Pro Leu
20 25 30
Phe Pro Gly Pro Ser Lys Pro Phe Trp Val Leu Val Val Val Gly Gly
35 40 45
Val Leu Ala Cys Tyr Ser Leu Leu Val Thr Val Ala Phe Ile Ile Phe
50 55 60
Trp Val
65
<210> 277
<211> 41
<212> Белок
<213> Homo sapiens
<220>
<223> CD28 (аминокислоты 180-220 P10747)
<400> 277
Arg Ser Lys Arg Ser Arg Leu Leu His Ser Asp Tyr Met Asn Met Thr
1 5 10 15
Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro
20 25 30
Pro Arg Asp Phe Ala Ala Tyr Arg Ser
35 40
<210> 278
<211> 41
<212> Белок
<213> Homo sapiens
<220>
<223> CD28 (LL - GG)
<400> 278
Arg Ser Lys Arg Ser Arg Gly Gly His Ser Asp Tyr Met Asn Met Thr
1 5 10 15
Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro
20 25 30
Pro Arg Asp Phe Ala Ala Tyr Arg Ser
35 40
<210> 279
<211> 42
<212> Белок
<213> Homo sapiens
<220>
<223> 4-1BB (аминокислоты 214-255 Q07011.1)
<400> 279
Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met
1 5 10 15
Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe
20 25 30
Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu
35 40
<210> 280
<211> 112
<212> Белок
<213> Homo sapiens
<220>
<223> CD3 дзета
<400> 280
Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly
1 5 10 15
Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr
20 25 30
Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys
35 40 45
Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys
50 55 60
Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg
65 70 75 80
Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala
85 90 95
Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
100 105 110
<210> 281
<211> 112
<212> Белок
<213> Homo sapiens
<220>
<223> CD3 дзета
<400> 281
Arg Val Lys Phe Ser Arg Ser Ala Glu Pro Pro Ala Tyr Gln Gln Gly
1 5 10 15
Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr
20 25 30
Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys
35 40 45
Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys
50 55 60
Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg
65 70 75 80
Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala
85 90 95
Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
100 105 110
<210> 282
<211> 112
<212> Белок
<213> Homo sapiens
<220>
<223> CD3 дзета
<400> 282
Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Lys Gln Gly
1 5 10 15
Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr
20 25 30
Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys
35 40 45
Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys
50 55 60
Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg
65 70 75 80
Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala
85 90 95
Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
100 105 110
<210> 283
<211> 252
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 12 / TCR 55 - (E7)11 - альфа нативный
<400> 283
Gly Glu Asp Val Glu Gln Ser Leu Phe Leu Ser Val Arg Glu Gly Asp
1 5 10 15
Ser Ser Val Ile Asn Cys Thr Tyr Thr Asp Ser Ser Ser Thr Tyr Leu
20 25 30
Tyr Trp Tyr Lys Gln Glu Pro Gly Ala Gly Leu Gln Leu Leu Thr Tyr
35 40 45
Ile Phe Ser Asn Met Asp Met Lys Gln Asp Gln Arg Leu Thr Val Leu
50 55 60
Leu Asn Lys Lys Asp Lys His Leu Ser Leu Arg Ile Ala Asp Thr Gln
65 70 75 80
Thr Gly Asp Ser Ala Ile Tyr Phe Cys Ala Val Pro Ser Gly Ala Thr
85 90 95
Asn Lys Leu Ile Phe Gly Thr Gly Thr Leu Leu Ala Val Gln Pro Asn
100 105 110
Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser
115 120 125
Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn
130 135 140
Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr Val
145 150 155 160
Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp
165 170 175
Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile
180 185 190
Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val
195 200 205
Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln
210 215 220
Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly
225 230 235 240
Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 284
<211> 252
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 12 / TCR 55- (E7)11 - альфа, цистеин-модифицированный
<400> 284
Gly Glu Asp Val Glu Gln Ser Leu Phe Leu Ser Val Arg Glu Gly Asp
1 5 10 15
Ser Ser Val Ile Asn Cys Thr Tyr Thr Asp Ser Ser Ser Thr Tyr Leu
20 25 30
Tyr Trp Tyr Lys Gln Glu Pro Gly Ala Gly Leu Gln Leu Leu Thr Tyr
35 40 45
Ile Phe Ser Asn Met Asp Met Lys Gln Asp Gln Arg Leu Thr Val Leu
50 55 60
Leu Asn Lys Lys Asp Lys His Leu Ser Leu Arg Ile Ala Asp Thr Gln
65 70 75 80
Thr Gly Asp Ser Ala Ile Tyr Phe Cys Ala Val Pro Ser Gly Ala Thr
85 90 95
Asn Lys Leu Ile Phe Gly Thr Gly Thr Leu Leu Ala Val Gln Pro Asn
100 105 110
Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser
115 120 125
Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn
130 135 140
Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys Val
145 150 155 160
Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp
165 170 175
Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile
180 185 190
Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val
195 200 205
Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln
210 215 220
Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly
225 230 235 240
Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 285
<211> 292
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 6, TCR 12, TCR 55 - (E7)11 - бета, нативный
<400> 285
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Ser Thr Thr
85 90 95
Arg Ser Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu
115 120 125
Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys
130 135 140
Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val
145 150 155 160
Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro Leu
165 170 175
Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg
180 185 190
Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg
195 200 205
Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln
210 215 220
Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly
225 230 235 240
Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu
245 250 255
Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr
260 265 270
Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys
275 280 285
Asp Ser Arg Gly
290
<210> 286
<211> 292
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 6, TCR 12, TCR 55 - (E7)11 - бета
цистеин-модифицированный
<400> 286
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Ser Thr Thr
85 90 95
Arg Ser Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu
115 120 125
Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys
130 135 140
Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val
145 150 155 160
Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro Leu
165 170 175
Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg
180 185 190
Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg
195 200 205
Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln
210 215 220
Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly
225 230 235 240
Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu
245 250 255
Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr
260 265 270
Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys
275 280 285
Asp Ser Arg Gly
290
<210> 287
<211> 251
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 13 - альфа нативный
<400> 287
Ala Gln Ser Val Thr Gln Leu Asp Ser His Val Ser Val Ser Glu Gly
1 5 10 15
Thr Pro Val Leu Leu Arg Cys Asn Tyr Ser Ser Ser Tyr Ser Pro Ser
20 25 30
Leu Phe Trp Tyr Val Gln His Pro Asn Lys Gly Leu Gln Leu Leu Leu
35 40 45
Lys Tyr Thr Ser Ala Ala Thr Leu Val Lys Gly Ile Asn Gly Phe Glu
50 55 60
Ala Glu Phe Lys Lys Ser Glu Thr Ser Phe His Leu Thr Lys Pro Ser
65 70 75 80
Ala His Met Ser Asp Ala Ala Glu Tyr Phe Cys Val Val Arg Gly Gly
85 90 95
Lys Leu Ile Phe Gly Gln Gly Thr Glu Leu Ser Val Lys Pro Asn Ile
100 105 110
Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser
115 120 125
Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val
130 135 140
Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr Val Leu
145 150 155 160
Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser
165 170 175
Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile
180 185 190
Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys
195 200 205
Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn
210 215 220
Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe
225 230 235 240
Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 288
<211> 251
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 13 - альфа, цистеин-модифицированный
<400> 288
Ala Gln Ser Val Thr Gln Leu Asp Ser His Val Ser Val Ser Glu Gly
1 5 10 15
Thr Pro Val Leu Leu Arg Cys Asn Tyr Ser Ser Ser Tyr Ser Pro Ser
20 25 30
Leu Phe Trp Tyr Val Gln His Pro Asn Lys Gly Leu Gln Leu Leu Leu
35 40 45
Lys Tyr Thr Ser Ala Ala Thr Leu Val Lys Gly Ile Asn Gly Phe Glu
50 55 60
Ala Glu Phe Lys Lys Ser Glu Thr Ser Phe His Leu Thr Lys Pro Ser
65 70 75 80
Ala His Met Ser Asp Ala Ala Glu Tyr Phe Cys Val Val Arg Gly Gly
85 90 95
Lys Leu Ile Phe Gly Gln Gly Thr Glu Leu Ser Val Lys Pro Asn Ile
100 105 110
Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser
115 120 125
Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val
130 135 140
Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys Val Leu
145 150 155 160
Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser
165 170 175
Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile
180 185 190
Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys
195 200 205
Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn
210 215 220
Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe
225 230 235 240
Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 289
<211> 293
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 13 - бета нативный
<400> 289
Gly Ala Gly Val Ser Gln Ser Pro Arg Tyr Lys Val Ala Lys Arg Gly
1 5 10 15
Gln Asp Val Ala Leu Arg Cys Asp Pro Ile Ser Gly His Val Ser Leu
20 25 30
Phe Trp Tyr Gln Gln Ala Leu Gly Gln Gly Pro Glu Phe Leu Thr Tyr
35 40 45
Phe Gln Asn Glu Ala Gln Leu Asp Lys Ser Gly Leu Pro Ser Asp Arg
50 55 60
Phe Phe Ala Glu Arg Pro Glu Gly Ser Val Ser Thr Leu Lys Ile Gln
65 70 75 80
Arg Thr Gln Gln Glu Asp Ser Ala Val Tyr Leu Cys Ala Ser Ser Pro
85 90 95
Thr Gly Thr Glu Arg Glu Leu Phe Phe Gly Glu Gly Ser Arg Leu Thr
100 105 110
Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe
115 120 125
Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val
130 135 140
Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp
145 150 155 160
Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro
165 170 175
Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser
180 185 190
Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe
195 200 205
Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr
210 215 220
Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp
225 230 235 240
Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val
245 250 255
Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu
260 265 270
Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg
275 280 285
Lys Asp Ser Arg Gly
290
<210> 290
<211> 293
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 13 -бета, цистеин-модифицированный
<400> 290
Gly Ala Gly Val Ser Gln Ser Pro Arg Tyr Lys Val Ala Lys Arg Gly
1 5 10 15
Gln Asp Val Ala Leu Arg Cys Asp Pro Ile Ser Gly His Val Ser Leu
20 25 30
Phe Trp Tyr Gln Gln Ala Leu Gly Gln Gly Pro Glu Phe Leu Thr Tyr
35 40 45
Phe Gln Asn Glu Ala Gln Leu Asp Lys Ser Gly Leu Pro Ser Asp Arg
50 55 60
Phe Phe Ala Glu Arg Pro Glu Gly Ser Val Ser Thr Leu Lys Ile Gln
65 70 75 80
Arg Thr Gln Gln Glu Asp Ser Ala Val Tyr Leu Cys Ala Ser Ser Pro
85 90 95
Thr Gly Thr Glu Arg Glu Leu Phe Phe Gly Glu Gly Ser Arg Leu Thr
100 105 110
Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe
115 120 125
Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val
130 135 140
Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp
145 150 155 160
Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro
165 170 175
Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser
180 185 190
Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe
195 200 205
Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr
210 215 220
Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp
225 230 235 240
Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val
245 250 255
Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu
260 265 270
Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg
275 280 285
Lys Asp Ser Arg Gly
290
<210> 291
<211> 252
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 14 -альфа нативный
<400> 291
Ile Leu Asn Val Glu Gln Ser Pro Gln Ser Leu His Val Gln Glu Gly
1 5 10 15
Asp Ser Thr Asn Phe Thr Cys Ser Phe Pro Ser Ser Asn Phe Tyr Ala
20 25 30
Leu His Trp Tyr Arg Trp Glu Thr Ala Lys Ser Pro Glu Ala Leu Phe
35 40 45
Val Met Thr Leu Asn Gly Asp Glu Lys Lys Lys Gly Arg Ile Ser Ala
50 55 60
Thr Leu Asn Thr Lys Glu Gly Tyr Ser Tyr Leu Tyr Ile Lys Gly Ser
65 70 75 80
Gln Pro Glu Asp Ser Ala Thr Tyr Leu Cys Ala Ser Gln Thr Gly Ala
85 90 95
Asn Asn Leu Phe Phe Gly Thr Gly Thr Arg Leu Thr Val Ile Pro Tyr
100 105 110
Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser
115 120 125
Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn
130 135 140
Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr Val
145 150 155 160
Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp
165 170 175
Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile
180 185 190
Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val
195 200 205
Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln
210 215 220
Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly
225 230 235 240
Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 292
<211> 252
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 14 -альфа, цистеин-модифицированный
<400> 292
Ile Leu Asn Val Glu Gln Ser Pro Gln Ser Leu His Val Gln Glu Gly
1 5 10 15
Asp Ser Thr Asn Phe Thr Cys Ser Phe Pro Ser Ser Asn Phe Tyr Ala
20 25 30
Leu His Trp Tyr Arg Trp Glu Thr Ala Lys Ser Pro Glu Ala Leu Phe
35 40 45
Val Met Thr Leu Asn Gly Asp Glu Lys Lys Lys Gly Arg Ile Ser Ala
50 55 60
Thr Leu Asn Thr Lys Glu Gly Tyr Ser Tyr Leu Tyr Ile Lys Gly Ser
65 70 75 80
Gln Pro Glu Asp Ser Ala Thr Tyr Leu Cys Ala Ser Gln Thr Gly Ala
85 90 95
Asn Asn Leu Phe Phe Gly Thr Gly Thr Arg Leu Thr Val Ile Pro Tyr
100 105 110
Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser
115 120 125
Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn
130 135 140
Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys Val
145 150 155 160
Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp
165 170 175
Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile
180 185 190
Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val
195 200 205
Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln
210 215 220
Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly
225 230 235 240
Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 293
<211> 291
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 14 - бета нативный
<400> 293
Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys Arg Thr Gly
1 5 10 15
Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His Glu Asn Met
20 25 30
Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu Ile Tyr Phe
35 40 45
Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro Glu Gly Tyr
50 55 60
Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile Leu Glu Ser
65 70 75 80
Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser Thr Phe Trp
85 90 95
Gly Gln Arg Arg Thr Glu Ala Phe Phe Gly Gln Gly Thr Arg Leu Thr
100 105 110
Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val Phe
115 120 125
Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val
130 135 140
Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp Trp
145 150 155 160
Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro
165 170 175
Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser
180 185 190
Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe
195 200 205
Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr
210 215 220
Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp
225 230 235 240
Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly Val
245 250 255
Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu
260 265 270
Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg
275 280 285
Lys Asp Phe
290
<210> 294
<211> 291
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 14 - бета, цистеин-модифицированный
<400> 294
Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys Arg Thr Gly
1 5 10 15
Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His Glu Asn Met
20 25 30
Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu Ile Tyr Phe
35 40 45
Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro Glu Gly Tyr
50 55 60
Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile Leu Glu Ser
65 70 75 80
Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser Thr Phe Trp
85 90 95
Gly Gln Arg Arg Thr Glu Ala Phe Phe Gly Gln Gly Thr Arg Leu Thr
100 105 110
Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val Phe
115 120 125
Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val
130 135 140
Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp Trp
145 150 155 160
Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro
165 170 175
Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser
180 185 190
Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe
195 200 205
Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr
210 215 220
Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp
225 230 235 240
Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly Val
245 250 255
Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu
260 265 270
Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg
275 280 285
Lys Asp Phe
290
<210> 295
<211> 111
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 12/ TCR 55 - альфа, вариабельная область
<400> 295
Gly Glu Asp Val Glu Gln Ser Leu Phe Leu Ser Val Arg Glu Gly Asp
1 5 10 15
Ser Ser Val Ile Asn Cys Thr Tyr Thr Asp Ser Ser Ser Thr Tyr Leu
20 25 30
Tyr Trp Tyr Lys Gln Glu Pro Gly Ala Gly Leu Gln Leu Leu Thr Tyr
35 40 45
Ile Phe Ser Asn Met Asp Met Lys Gln Asp Gln Arg Leu Thr Val Leu
50 55 60
Leu Asn Lys Lys Asp Lys His Leu Ser Leu Arg Ile Ala Asp Thr Gln
65 70 75 80
Thr Gly Asp Ser Ala Ile Tyr Phe Cys Ala Val Pro Ser Gly Ala Thr
85 90 95
Asn Lys Leu Ile Phe Gly Thr Gly Thr Leu Leu Ala Val Gln Pro
100 105 110
<210> 296
<211> 113
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 6, TCR 12, TCR 55 - бета, вариабельная область
<400> 296
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Ser Thr Thr
85 90 95
Arg Ser Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Thr
<210> 297
<211> 110
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 13 - альфа, вариабельная область
<400> 297
Ala Gln Ser Val Thr Gln Leu Asp Ser His Val Ser Val Ser Glu Gly
1 5 10 15
Thr Pro Val Leu Leu Arg Cys Asn Tyr Ser Ser Ser Tyr Ser Pro Ser
20 25 30
Leu Phe Trp Tyr Val Gln His Pro Asn Lys Gly Leu Gln Leu Leu Leu
35 40 45
Lys Tyr Thr Ser Ala Ala Thr Leu Val Lys Gly Ile Asn Gly Phe Glu
50 55 60
Ala Glu Phe Lys Lys Ser Glu Thr Ser Phe His Leu Thr Lys Pro Ser
65 70 75 80
Ala His Met Ser Asp Ala Ala Glu Tyr Phe Cys Val Val Arg Gly Gly
85 90 95
Lys Leu Ile Phe Gly Gln Gly Thr Glu Leu Ser Val Lys Pro
100 105 110
<210> 298
<211> 114
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 13 - бета, вариабельная область
<400> 298
Gly Ala Gly Val Ser Gln Ser Pro Arg Tyr Lys Val Ala Lys Arg Gly
1 5 10 15
Gln Asp Val Ala Leu Arg Cys Asp Pro Ile Ser Gly His Val Ser Leu
20 25 30
Phe Trp Tyr Gln Gln Ala Leu Gly Gln Gly Pro Glu Phe Leu Thr Tyr
35 40 45
Phe Gln Asn Glu Ala Gln Leu Asp Lys Ser Gly Leu Pro Ser Asp Arg
50 55 60
Phe Phe Ala Glu Arg Pro Glu Gly Ser Val Ser Thr Leu Lys Ile Gln
65 70 75 80
Arg Thr Gln Gln Glu Asp Ser Ala Val Tyr Leu Cys Ala Ser Ser Pro
85 90 95
Thr Gly Thr Glu Arg Glu Leu Phe Phe Gly Glu Gly Ser Arg Leu Thr
100 105 110
Val Leu
<210> 299
<211> 111
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 14 - альфа, вариабельная область
<400> 299
Ile Leu Asn Val Glu Gln Ser Pro Gln Ser Leu His Val Gln Glu Gly
1 5 10 15
Asp Ser Thr Asn Phe Thr Cys Ser Phe Pro Ser Ser Asn Phe Tyr Ala
20 25 30
Leu His Trp Tyr Arg Trp Glu Thr Ala Lys Ser Pro Glu Ala Leu Phe
35 40 45
Val Met Thr Leu Asn Gly Asp Glu Lys Lys Lys Gly Arg Ile Ser Ala
50 55 60
Thr Leu Asn Thr Lys Glu Gly Tyr Ser Tyr Leu Tyr Ile Lys Gly Ser
65 70 75 80
Gln Pro Glu Asp Ser Ala Thr Tyr Leu Cys Ala Ser Gln Thr Gly Ala
85 90 95
Asn Asn Leu Phe Phe Gly Thr Gly Thr Arg Leu Thr Val Ile Pro
100 105 110
<210> 300
<211> 114
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 14 - бета, вариабельная область
<400> 300
Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys Arg Thr Gly
1 5 10 15
Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His Glu Asn Met
20 25 30
Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu Ile Tyr Phe
35 40 45
Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro Glu Gly Tyr
50 55 60
Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile Leu Glu Ser
65 70 75 80
Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser Thr Phe Trp
85 90 95
Gly Gln Arg Arg Thr Glu Ala Phe Phe Gly Gln Gly Thr Arg Leu Thr
100 105 110
Val Val
<210> 301
<211> 11
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 12/ TCR 55 CDR3 альфа
<400> 301
Ala Val Pro Ser Gly Ala Thr Asn Lys Leu Ile
1 5 10
<210> 302
<211> 6
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 13 CDR1 альфа
<400> 302
Ser Ser Tyr Ser Pro Ser
1 5
<210> 303
<211> 8
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 13 CDR2 альфа
<400> 303
Tyr Thr Ser Ala Ala Thr Leu Val
1 5
<210> 304
<211> 8
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 13 CDR3 альфа
<400> 304
Val Val Arg Gly Gly Lys Leu Ile
1 5
<210> 305
<211> 12
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 13 CDR3 бета
<400> 305
Ala Ser Ser Pro Thr Gly Thr Glu Arg Glu Leu Phe
1 5 10
<210> 306
<211> 6
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 14 CDR1 альфа
<400> 306
Ser Ser Asn Phe Tyr Ala
1 5
<210> 307
<211> 7
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 14 CDR2 альфа
<400> 307
Met Thr Leu Asn Gly Asp Glu
1 5
<210> 308
<211> 10
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 14 CDR3 альфа
<400> 308
Ala Ser Gln Thr Gly Ala Asn Asn Leu Phe
1 5 10
<210> 309
<211> 13
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 14 CDR3 бета
<400> 309
Ala Ser Thr Phe Trp Gly Gln Arg Arg Thr Glu Ala Phe
1 5 10
<210> 310
<211> 19
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 13 альфа, Сигнальная последовательность
<400> 310
Met Leu Leu Leu Leu Val Pro Val Leu Glu Val Ile Phe Thr Leu Gly
1 5 10 15
Gly Thr Arg
<210> 311
<211> 22
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 14 альфа, Сигнальная последовательность
<400> 311
Met Glu Lys Asn Pro Leu Ala Ala Pro Leu Leu Ile Leu Trp Phe His
1 5 10 15
Leu Asp Cys Val Ser Ser
20
<210> 312
<211> 312
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 13 - бета, цистеин-модифицированный
<400> 312
Met Gly Thr Arg Leu Leu Cys Trp Val Val Leu Gly Phe Leu Gly Thr
1 5 10 15
Asp His Thr Gly Ala Gly Val Ser Gln Ser Pro Arg Tyr Lys Val Ala
20 25 30
Lys Arg Gly Gln Asp Val Ala Leu Arg Cys Asp Pro Ile Ser Gly His
35 40 45
Val Ser Leu Phe Trp Tyr Gln Gln Ala Leu Gly Gln Gly Pro Glu Phe
50 55 60
Leu Thr Tyr Phe Gln Asn Glu Ala Gln Leu Asp Lys Ser Gly Leu Pro
65 70 75 80
Ser Asp Arg Phe Phe Ala Glu Arg Pro Glu Gly Ser Val Ser Thr Leu
85 90 95
Lys Ile Gln Arg Thr Gln Gln Glu Asp Ser Ala Val Tyr Leu Cys Ala
100 105 110
Ser Ser Pro Thr Gly Thr Glu Arg Glu Leu Phe Phe Gly Glu Gly Ser
115 120 125
Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 313
<211> 274
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 14 - альфа нативный
<400> 313
Met Glu Lys Asn Pro Leu Ala Ala Pro Leu Leu Ile Leu Trp Phe His
1 5 10 15
Leu Asp Cys Val Ser Ser Ile Leu Asn Val Glu Gln Ser Pro Gln Ser
20 25 30
Leu His Val Gln Glu Gly Asp Ser Thr Asn Phe Thr Cys Ser Phe Pro
35 40 45
Ser Ser Asn Phe Tyr Ala Leu His Trp Tyr Arg Trp Glu Thr Ala Lys
50 55 60
Ser Pro Glu Ala Leu Phe Val Met Thr Leu Asn Gly Asp Glu Lys Lys
65 70 75 80
Lys Gly Arg Ile Ser Ala Thr Leu Asn Thr Lys Glu Gly Tyr Ser Tyr
85 90 95
Leu Tyr Ile Lys Gly Ser Gln Pro Glu Asp Ser Ala Thr Tyr Leu Cys
100 105 110
Ala Ser Gln Thr Gly Ala Asn Asn Leu Phe Phe Gly Thr Gly Thr Arg
115 120 125
Leu Thr Val Ile Pro Tyr Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln
130 135 140
Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp
145 150 155 160
Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr
165 170 175
Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser
180 185 190
Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn
195 200 205
Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro
210 215 220
Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp
225 230 235 240
Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu
245 250 255
Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp
260 265 270
Ser Ser
<210> 314
<211> 274
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 14 - альфа, цистеин-модифицированный
<400> 314
Met Glu Lys Asn Pro Leu Ala Ala Pro Leu Leu Ile Leu Trp Phe His
1 5 10 15
Leu Asp Cys Val Ser Ser Ile Leu Asn Val Glu Gln Ser Pro Gln Ser
20 25 30
Leu His Val Gln Glu Gly Asp Ser Thr Asn Phe Thr Cys Ser Phe Pro
35 40 45
Ser Ser Asn Phe Tyr Ala Leu His Trp Tyr Arg Trp Glu Thr Ala Lys
50 55 60
Ser Pro Glu Ala Leu Phe Val Met Thr Leu Asn Gly Asp Glu Lys Lys
65 70 75 80
Lys Gly Arg Ile Ser Ala Thr Leu Asn Thr Lys Glu Gly Tyr Ser Tyr
85 90 95
Leu Tyr Ile Lys Gly Ser Gln Pro Glu Asp Ser Ala Thr Tyr Leu Cys
100 105 110
Ala Ser Gln Thr Gly Ala Asn Asn Leu Phe Phe Gly Thr Gly Thr Arg
115 120 125
Leu Thr Val Ile Pro Tyr Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln
130 135 140
Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp
145 150 155 160
Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr
165 170 175
Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser
180 185 190
Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn
195 200 205
Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro
210 215 220
Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp
225 230 235 240
Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu
245 250 255
Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp
260 265 270
Ser Ser
<210> 315
<211> 310
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 14 - бета нативный
<400> 315
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Thr Phe Trp Gly Gln Arg Arg Thr Glu Ala Phe Phe Gly Gln Gly Thr
115 120 125
Arg Leu Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe
305 310
<210> 316
<211> 310
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 14 - бета, цистеин-модифицированный
<400> 316
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Thr Phe Trp Gly Gln Arg Arg Thr Glu Ala Phe Phe Gly Gln Gly Thr
115 120 125
Arg Leu Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe
305 310
<210> 317
<211> 137
<212> Белок
<213> Mus musculus
<220>
<223> Последовательность мышиной константной области альфа
<400> 317
Asn Ile Gln Asn Pro Glu Pro Ala Val Tyr Gln Leu Lys Asp Pro Arg
1 5 10 15
Ser Gln Asp Ser Thr Leu Cys Leu Phe Thr Asp Phe Asp Ser Gln Ile
20 25 30
Asn Val Pro Lys Thr Met Glu Ser Gly Thr Phe Ile Thr Asp Lys Thr
35 40 45
Val Leu Asp Met Lys Ala Met Asp Ser Lys Ser Asn Gly Ala Ile Ala
50 55 60
Trp Ser Asn Gln Thr Ser Phe Thr Cys Gln Asp Ile Phe Lys Glu Thr
65 70 75 80
Asn Ala Thr Tyr Pro Ser Ser Asp Val Pro Cys Asp Ala Thr Leu Thr
85 90 95
Glu Lys Ser Phe Glu Thr Asp Met Asn Leu Asn Phe Gln Asn Leu Ser
100 105 110
Val Met Gly Leu Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu
115 120 125
Leu Met Thr Leu Arg Leu Trp Ser Ser
130 135
<210> 318
<211> 275
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 3 - альфа нативный
<400> 318
Met Ser Leu Ser Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu
1 5 10 15
Gly Pro Gly Ile Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe
20 25 30
Val Gln Glu Lys Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser
35 40 45
Asp Gln Ser Tyr Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu
50 55 60
Met Ile Phe Leu Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr
65 70 75 80
Glu Gly Arg Tyr Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn
85 90 95
Leu Val Ile Ser Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys
100 105 110
Ala Met Arg Glu Gly Arg Gly Phe Lys Thr Ile Phe Gly Ala Gly Thr
115 120 125
Arg Leu Phe Val Lys Ala Asn Ile Gln Lys Pro Asp Pro Ala Val Tyr
130 135 140
Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr
145 150 155 160
Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val
165 170 175
Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys
180 185 190
Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala
195 200 205
Asn Ala Phe Asn Asn Ser Ile Ile Pro Ala Asp Thr Phe Phe Pro Ser
210 215 220
Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr
225 230 235 240
Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile
245 250 255
Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu
260 265 270
Trp Ser Ser
275
<210> 319
<211> 275
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 3 - альфа, цистеин-модифицированный
<400> 319
Met Ser Leu Ser Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu
1 5 10 15
Gly Pro Gly Ile Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe
20 25 30
Val Gln Glu Lys Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser
35 40 45
Asp Gln Ser Tyr Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu
50 55 60
Met Ile Phe Leu Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr
65 70 75 80
Glu Gly Arg Tyr Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn
85 90 95
Leu Val Ile Ser Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys
100 105 110
Ala Met Arg Glu Gly Arg Gly Phe Lys Thr Ile Phe Gly Ala Gly Thr
115 120 125
Arg Leu Phe Val Lys Ala Asn Ile Gln Lys Pro Asp Pro Ala Val Tyr
130 135 140
Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr
145 150 155 160
Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val
165 170 175
Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys
180 185 190
Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala
195 200 205
Asn Ala Phe Asn Asn Ser Ile Ile Pro Ala Asp Thr Phe Phe Pro Ser
210 215 220
Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr
225 230 235 240
Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile
245 250 255
Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu
260 265 270
Trp Ser Ser
275
<210> 320
<211> 313
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 3 - бета нативный
<400> 320
Met Gly Thr Arg Leu Leu Cys Trp Val Val Leu Gly Phe Leu Gly Thr
1 5 10 15
Asp His Thr Gly Ala Gly Val Ser Gln Ser Pro Arg Tyr Lys Val Ala
20 25 30
Lys Arg Gly Gln Asp Val Ala Leu Arg Cys Asp Pro Ile Ser Gly His
35 40 45
Val Ser Leu Phe Trp Tyr Gln Gln Ala Leu Gly Gln Gly Pro Glu Phe
50 55 60
Leu Thr Tyr Phe Gln Asn Glu Ala Gln Leu Asp Lys Ser Gly Leu Pro
65 70 75 80
Ser Asp Arg Phe Phe Ala Glu Arg Pro Glu Gly Ser Val Ser Thr Leu
85 90 95
Lys Ile Gln Arg Thr Gln Gln Glu Asp Ser Ala Val Tyr Leu Cys Ala
100 105 110
Ser Ser His Leu Ala Gly Phe Thr Gly Glu Leu Phe Phe Gly Glu Gly
115 120 125
Ser Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu
130 135 140
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
145 150 155 160
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu
165 170 175
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr
180 185 190
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
195 200 205
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
210 215 220
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
225 230 235 240
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
245 250 255
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr
260 265 270
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
275 280 285
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
290 295 300
Met Val Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 321
<211> 313
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 3 - бета, цистеин-модифицированный
<400> 321
Met Gly Thr Arg Leu Leu Cys Trp Val Val Leu Gly Phe Leu Gly Thr
1 5 10 15
Asp His Thr Gly Ala Gly Val Ser Gln Ser Pro Arg Tyr Lys Val Ala
20 25 30
Lys Arg Gly Gln Asp Val Ala Leu Arg Cys Asp Pro Ile Ser Gly His
35 40 45
Val Ser Leu Phe Trp Tyr Gln Gln Ala Leu Gly Gln Gly Pro Glu Phe
50 55 60
Leu Thr Tyr Phe Gln Asn Glu Ala Gln Leu Asp Lys Ser Gly Leu Pro
65 70 75 80
Ser Asp Arg Phe Phe Ala Glu Arg Pro Glu Gly Ser Val Ser Thr Leu
85 90 95
Lys Ile Gln Arg Thr Gln Gln Glu Asp Ser Ala Val Tyr Leu Cys Ala
100 105 110
Ser Ser His Leu Ala Gly Phe Thr Gly Glu Leu Phe Phe Gly Glu Gly
115 120 125
Ser Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu
130 135 140
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
145 150 155 160
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu
165 170 175
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr
180 185 190
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
195 200 205
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
210 215 220
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
225 230 235 240
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
245 250 255
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr
260 265 270
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
275 280 285
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
290 295 300
Met Val Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 322
<211> 271
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 4 - (E6)29 альфа нативный
<400> 322
Met Lys Leu Val Thr Ser Ile Thr Val Leu Leu Ser Leu Gly Ile Met
1 5 10 15
Gly Asp Ala Lys Thr Thr Gln Pro Asn Ser Met Glu Ser Asn Glu Glu
20 25 30
Glu Pro Val His Leu Pro Cys Asn His Ser Thr Ile Ser Gly Thr Asp
35 40 45
Tyr Ile His Trp Tyr Arg Gln Leu Pro Ser Gln Gly Pro Glu Tyr Val
50 55 60
Ile His Gly Leu Thr Ser Asn Val Asn Asn Arg Met Ala Ser Leu Ala
65 70 75 80
Ile Ala Glu Asp Arg Lys Ser Ser Thr Leu Ile Leu His Arg Ala Thr
85 90 95
Leu Arg Asp Ala Ala Val Tyr Tyr Cys Ile Leu Leu Val Ile Arg Gly
100 105 110
Thr Ser Tyr Gly Lys Leu Thr Phe Gly Gln Gly Thr Ile Leu Thr Val
115 120 125
His Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp
130 135 140
Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser
145 150 155 160
Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp
165 170 175
Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala
180 185 190
Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn
195 200 205
Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser
210 215 220
Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu
225 230 235 240
Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys
245 250 255
Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
260 265 270
<210> 323
<211> 271
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 4 - (E6)29 альфа, цистеин-модифицированный
<400> 323
Met Lys Leu Val Thr Ser Ile Thr Val Leu Leu Ser Leu Gly Ile Met
1 5 10 15
Gly Asp Ala Lys Thr Thr Gln Pro Asn Ser Met Glu Ser Asn Glu Glu
20 25 30
Glu Pro Val His Leu Pro Cys Asn His Ser Thr Ile Ser Gly Thr Asp
35 40 45
Tyr Ile His Trp Tyr Arg Gln Leu Pro Ser Gln Gly Pro Glu Tyr Val
50 55 60
Ile His Gly Leu Thr Ser Asn Val Asn Asn Arg Met Ala Ser Leu Ala
65 70 75 80
Ile Ala Glu Asp Arg Lys Ser Ser Thr Leu Ile Leu His Arg Ala Thr
85 90 95
Leu Arg Asp Ala Ala Val Tyr Tyr Cys Ile Leu Leu Val Ile Arg Gly
100 105 110
Thr Ser Tyr Gly Lys Leu Thr Phe Gly Gln Gly Thr Ile Leu Thr Val
115 120 125
His Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp
130 135 140
Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser
145 150 155 160
Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp
165 170 175
Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala
180 185 190
Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn
195 200 205
Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser
210 215 220
Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu
225 230 235 240
Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys
245 250 255
Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
260 265 270
<210> 324
<211> 310
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 4 - (E6)29 бета нативный
<400> 324
Met Gly Thr Ser Leu Leu Cys Trp Met Ala Leu Cys Leu Leu Gly Ala
1 5 10 15
Asp His Ala Asp Thr Gly Val Ser Gln Asp Pro Arg His Lys Ile Thr
20 25 30
Lys Arg Gly Gln Asn Val Thr Phe Arg Cys Asp Pro Ile Ser Glu His
35 40 45
Asn Arg Leu Tyr Trp Tyr Arg Gln Thr Leu Gly Gln Gly Pro Glu Phe
50 55 60
Leu Thr Tyr Phe Gln Asn Glu Ala Gln Leu Glu Lys Ser Arg Leu Leu
65 70 75 80
Ser Asp Arg Phe Ser Ala Glu Arg Pro Lys Gly Ser Phe Ser Thr Leu
85 90 95
Glu Ile Gln Arg Thr Glu Gln Gly Asp Ser Ala Met Tyr Leu Cys Ala
100 105 110
Ser Ser Pro Gly Gly Gly Asn Thr Glu Ala Phe Phe Gly Gln Gly Thr
115 120 125
Arg Leu Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe
305 310
<210> 325
<211> 310
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 4 - (E6)29 бета, цистеин-модифицированный
<400> 325
Met Gly Thr Ser Leu Leu Cys Trp Met Ala Leu Cys Leu Leu Gly Ala
1 5 10 15
Asp His Ala Asp Thr Gly Val Ser Gln Asp Pro Arg His Lys Ile Thr
20 25 30
Lys Arg Gly Gln Asn Val Thr Phe Arg Cys Asp Pro Ile Ser Glu His
35 40 45
Asn Arg Leu Tyr Trp Tyr Arg Gln Thr Leu Gly Gln Gly Pro Glu Phe
50 55 60
Leu Thr Tyr Phe Gln Asn Glu Ala Gln Leu Glu Lys Ser Arg Leu Leu
65 70 75 80
Ser Asp Arg Phe Ser Ala Glu Arg Pro Lys Gly Ser Phe Ser Thr Leu
85 90 95
Glu Ile Gln Arg Thr Glu Gln Gly Asp Ser Ala Met Tyr Leu Cys Ala
100 105 110
Ser Ser Pro Gly Gly Gly Asn Thr Glu Ala Phe Phe Gly Gln Gly Thr
115 120 125
Arg Leu Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe
305 310
<210> 326
<211> 278
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 5 - (E6)29 - TCR альфа нативный
<400> 326
Met Ser Leu Ser Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu
1 5 10 15
Gly Pro Gly Ile Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe
20 25 30
Val Gln Glu Lys Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser
35 40 45
Asp Gln Ser Tyr Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu
50 55 60
Met Ile Phe Leu Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr
65 70 75 80
Glu Gly Arg Tyr Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn
85 90 95
Leu Val Ile Ser Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys
100 105 110
Ala Met Arg Glu Gly Thr Gly Thr Ser Tyr Gly Lys Leu Thr Phe Gly
115 120 125
Gln Gly Thr Ile Leu Thr Val His Pro Asn Ile Gln Asn Pro Asp Pro
130 135 140
Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys
145 150 155 160
Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp
165 170 175
Ser Asp Val Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met
180 185 190
Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe
195 200 205
Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe
210 215 220
Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser
225 230 235 240
Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly
245 250 255
Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr
260 265 270
Leu Arg Leu Trp Ser Ser
275
<210> 327
<211> 278
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 5 - (E6)29 - TCR альфа, цистеин-модифицированный
<400> 327
Met Ser Leu Ser Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu
1 5 10 15
Gly Pro Gly Ile Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe
20 25 30
Val Gln Glu Lys Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser
35 40 45
Asp Gln Ser Tyr Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu
50 55 60
Met Ile Phe Leu Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr
65 70 75 80
Glu Gly Arg Tyr Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn
85 90 95
Leu Val Ile Ser Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys
100 105 110
Ala Met Arg Glu Gly Thr Gly Thr Ser Tyr Gly Lys Leu Thr Phe Gly
115 120 125
Gln Gly Thr Ile Leu Thr Val His Pro Asn Ile Gln Asn Pro Asp Pro
130 135 140
Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys
145 150 155 160
Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp
165 170 175
Ser Asp Val Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met
180 185 190
Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe
195 200 205
Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe
210 215 220
Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser
225 230 235 240
Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly
245 250 255
Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr
260 265 270
Leu Arg Leu Trp Ser Ser
275
<210> 328
<211> 310
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 5 - (E6)29 - TCR бета нативный
<400> 328
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Ser Pro Trp Gly Glu Thr His Gln Pro Gln His Phe Gly Asp Gly Thr
115 120 125
Arg Leu Ser Ile Leu Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe
305 310
<210> 329
<211> 310
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 5 - (E6)29 - TCR бета, цистеин-модифицированный
<400> 329
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Ser Pro Trp Gly Glu Thr His Gln Pro Gln His Phe Gly Asp Gly Thr
115 120 125
Arg Leu Ser Ile Leu Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe
305 310
<210> 330
<211> 274
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 6 - альфа нативный
<400> 330
Met Lys Thr Phe Ala Gly Phe Ser Phe Leu Phe Leu Trp Leu Gln Leu
1 5 10 15
Asp Cys Met Ser Arg Gly Glu Asp Val Glu Gln Ser Leu Phe Leu Ser
20 25 30
Val Arg Glu Gly Asp Ser Ser Val Ile Asn Cys Thr Tyr Thr Asp Ser
35 40 45
Ser Ser Thr Tyr Leu Tyr Trp Tyr Lys Gln Glu Pro Gly Ala Gly Leu
50 55 60
Gln Leu Leu Thr Tyr Ile Phe Ser Asn Met Asp Met Lys Gln Asp Gln
65 70 75 80
Arg Leu Thr Val Leu Leu Asn Lys Lys Asp Lys His Leu Ser Leu Arg
85 90 95
Ile Ala Asp Thr Gln Thr Gly Asp Ser Ala Ile Tyr Phe Cys Ala Glu
100 105 110
Ser Ile Arg Gly Phe Gly Asn Val Leu His Cys Gly Ser Gly Thr Gln
115 120 125
Val Ile Val Leu Pro His Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln
130 135 140
Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp
145 150 155 160
Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr
165 170 175
Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser
180 185 190
Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn
195 200 205
Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro
210 215 220
Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp
225 230 235 240
Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu
245 250 255
Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp
260 265 270
Ser Ser
<210> 331
<211> 274
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 6 - альфа, цистеин-модифицированный
<400> 331
Met Lys Thr Phe Ala Gly Phe Ser Phe Leu Phe Leu Trp Leu Gln Leu
1 5 10 15
Asp Cys Met Ser Arg Gly Glu Asp Val Glu Gln Ser Leu Phe Leu Ser
20 25 30
Val Arg Glu Gly Asp Ser Ser Val Ile Asn Cys Thr Tyr Thr Asp Ser
35 40 45
Ser Ser Thr Tyr Leu Tyr Trp Tyr Lys Gln Glu Pro Gly Ala Gly Leu
50 55 60
Gln Leu Leu Thr Tyr Ile Phe Ser Asn Met Asp Met Lys Gln Asp Gln
65 70 75 80
Arg Leu Thr Val Leu Leu Asn Lys Lys Asp Lys His Leu Ser Leu Arg
85 90 95
Ile Ala Asp Thr Gln Thr Gly Asp Ser Ala Ile Tyr Phe Cys Ala Glu
100 105 110
Ser Ile Arg Gly Phe Gly Asn Val Leu His Cys Gly Ser Gly Thr Gln
115 120 125
Val Ile Val Leu Pro His Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln
130 135 140
Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp
145 150 155 160
Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr
165 170 175
Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser
180 185 190
Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn
195 200 205
Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro
210 215 220
Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp
225 230 235 240
Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu
245 250 255
Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp
260 265 270
Ser Ser
<210> 332
<211> 311
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 6, TCR 12 - бета нативный
<400> 332
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ser Thr Thr Arg Ser Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 333
<211> 311
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 6, TCR 12 - бета, цистеин-модифицированный
<400> 333
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ser Thr Thr Arg Ser Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 334
<211> 275
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 7/ TCR 54 - (E7)11 - альфа нативный
<400> 334
Met Lys Lys His Leu Thr Thr Phe Leu Val Ile Leu Trp Leu Tyr Phe
1 5 10 15
Tyr Arg Gly Asn Gly Lys Asn Gln Val Glu Gln Ser Pro Gln Ser Leu
20 25 30
Ile Ile Leu Glu Gly Lys Asn Cys Thr Leu Gln Cys Asn Tyr Thr Val
35 40 45
Ser Pro Phe Ser Asn Leu Arg Trp Tyr Lys Gln Asp Thr Gly Arg Gly
50 55 60
Pro Val Ser Leu Thr Ile Met Thr Phe Ser Glu Asn Thr Lys Ser Asn
65 70 75 80
Gly Arg Tyr Thr Ala Thr Leu Asp Ala Asp Thr Lys Gln Ser Ser Leu
85 90 95
His Ile Thr Ala Ser Gln Leu Ser Asp Ser Ala Ser Tyr Ile Cys Val
100 105 110
Val Ser Arg Asp Asn Tyr Gly Gln Asn Phe Val Phe Gly Pro Gly Thr
115 120 125
Arg Leu Ser Val Leu Pro Tyr Ile Gln Asn Pro Asp Pro Ala Val Tyr
130 135 140
Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr
145 150 155 160
Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val
165 170 175
Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys
180 185 190
Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala
195 200 205
Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser
210 215 220
Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr
225 230 235 240
Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile
245 250 255
Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu
260 265 270
Trp Ser Ser
275
<210> 335
<211> 275
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 7/ TCR 54 - (E7)11 - альфа, цистеин-модифицированный
<400> 335
Met Lys Lys His Leu Thr Thr Phe Leu Val Ile Leu Trp Leu Tyr Phe
1 5 10 15
Tyr Arg Gly Asn Gly Lys Asn Gln Val Glu Gln Ser Pro Gln Ser Leu
20 25 30
Ile Ile Leu Glu Gly Lys Asn Cys Thr Leu Gln Cys Asn Tyr Thr Val
35 40 45
Ser Pro Phe Ser Asn Leu Arg Trp Tyr Lys Gln Asp Thr Gly Arg Gly
50 55 60
Pro Val Ser Leu Thr Ile Met Thr Phe Ser Glu Asn Thr Lys Ser Asn
65 70 75 80
Gly Arg Tyr Thr Ala Thr Leu Asp Ala Asp Thr Lys Gln Ser Ser Leu
85 90 95
His Ile Thr Ala Ser Gln Leu Ser Asp Ser Ala Ser Tyr Ile Cys Val
100 105 110
Val Ser Arg Asp Asn Tyr Gly Gln Asn Phe Val Phe Gly Pro Gly Thr
115 120 125
Arg Leu Ser Val Leu Pro Tyr Ile Gln Asn Pro Asp Pro Ala Val Tyr
130 135 140
Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr
145 150 155 160
Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val
165 170 175
Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys
180 185 190
Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala
195 200 205
Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser
210 215 220
Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr
225 230 235 240
Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile
245 250 255
Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu
260 265 270
Trp Ser Ser
275
<210> 336
<211> 309
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 7/ TCR 54 -(E7)11 -бета нативный
<400> 336
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ile Thr Asp Arg Thr Asn Tyr Gly Tyr Thr Phe Gly Ser Gly Thr Arg
115 120 125
Leu Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Phe
305
<210> 337
<211> 309
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 7/ TCR 54 - (E7)11 -бета, цистеин-модифицированный
<400> 337
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ile Thr Asp Arg Thr Asn Tyr Gly Tyr Thr Phe Gly Ser Gly Thr Arg
115 120 125
Leu Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Phe
305
<210> 338
<211> 272
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 8 - альфа нативный
<400> 338
Met Glu Thr Leu Leu Gly Leu Leu Ile Leu Trp Leu Gln Leu Gln Trp
1 5 10 15
Val Ser Ser Lys Gln Glu Val Thr Gln Ile Pro Ala Ala Leu Ser Val
20 25 30
Pro Glu Gly Glu Asn Leu Val Leu Asn Cys Ser Phe Thr Asp Ser Ala
35 40 45
Ile Tyr Asn Leu Gln Trp Phe Arg Gln Asp Pro Gly Lys Gly Leu Thr
50 55 60
Ser Leu Leu Leu Ile Gln Ser Ser Gln Arg Glu Gln Thr Ser Gly Arg
65 70 75 80
Leu Asn Ala Ser Leu Asp Lys Ser Ser Gly Arg Ser Thr Leu Tyr Ile
85 90 95
Ala Ala Ser Gln Pro Gly Asp Ser Ala Thr Tyr Leu Cys Ala Val Arg
100 105 110
Pro Leu Gly Asn Thr Pro Leu Val Phe Gly Lys Gly Thr Arg Leu Ser
115 120 125
Val Ile Ala Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg
130 135 140
Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp
145 150 155 160
Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr
165 170 175
Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser
180 185 190
Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe
195 200 205
Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser
210 215 220
Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn
225 230 235 240
Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu
245 250 255
Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
260 265 270
<210> 339
<211> 272
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 8 - альфа, цистеин-модифицированный
<400> 339
Met Glu Thr Leu Leu Gly Leu Leu Ile Leu Trp Leu Gln Leu Gln Trp
1 5 10 15
Val Ser Ser Lys Gln Glu Val Thr Gln Ile Pro Ala Ala Leu Ser Val
20 25 30
Pro Glu Gly Glu Asn Leu Val Leu Asn Cys Ser Phe Thr Asp Ser Ala
35 40 45
Ile Tyr Asn Leu Gln Trp Phe Arg Gln Asp Pro Gly Lys Gly Leu Thr
50 55 60
Ser Leu Leu Leu Ile Gln Ser Ser Gln Arg Glu Gln Thr Ser Gly Arg
65 70 75 80
Leu Asn Ala Ser Leu Asp Lys Ser Ser Gly Arg Ser Thr Leu Tyr Ile
85 90 95
Ala Ala Ser Gln Pro Gly Asp Ser Ala Thr Tyr Leu Cys Ala Val Arg
100 105 110
Pro Leu Gly Asn Thr Pro Leu Val Phe Gly Lys Gly Thr Arg Leu Ser
115 120 125
Val Ile Ala Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg
130 135 140
Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp
145 150 155 160
Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr
165 170 175
Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser
180 185 190
Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe
195 200 205
Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser
210 215 220
Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn
225 230 235 240
Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu
245 250 255
Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
260 265 270
<210> 340
<211> 606
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 12/ TCR 55 полноразмерная последовательность, нативный
<400> 340
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ser Thr Thr Arg Ser Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser Leu
305 310 315 320
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Lys Thr
325 330 335
Phe Ala Gly Phe Ser Phe Leu Phe Leu Trp Leu Gln Leu Asp Cys Met
340 345 350
Ser Arg Gly Glu Asp Val Glu Gln Ser Leu Phe Leu Ser Val Arg Glu
355 360 365
Gly Asp Ser Ser Val Ile Asn Cys Thr Tyr Thr Asp Ser Ser Ser Thr
370 375 380
Tyr Leu Tyr Trp Tyr Lys Gln Glu Pro Gly Ala Gly Leu Gln Leu Leu
385 390 395 400
Thr Tyr Ile Phe Ser Asn Met Asp Met Lys Gln Asp Gln Arg Leu Thr
405 410 415
Val Leu Leu Asn Lys Lys Asp Lys His Leu Ser Leu Arg Ile Ala Asp
420 425 430
Thr Gln Thr Gly Asp Ser Ala Ile Tyr Phe Cys Ala Val Pro Ser Gly
435 440 445
Ala Thr Asn Lys Leu Ile Phe Gly Thr Gly Thr Leu Leu Ala Val Gln
450 455 460
Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser
465 470 475 480
Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln
485 490 495
Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys
500 505 510
Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val
515 520 525
Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn
530 535 540
Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys
545 550 555 560
Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn
565 570 575
Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val
580 585 590
Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600 605
<210> 341
<211> 604
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 13 полноразмерная последовательность, нативный
<400> 341
Met Gly Thr Arg Leu Leu Cys Trp Val Val Leu Gly Phe Leu Gly Thr
1 5 10 15
Asp His Thr Gly Ala Gly Val Ser Gln Ser Pro Arg Tyr Lys Val Ala
20 25 30
Lys Arg Gly Gln Asp Val Ala Leu Arg Cys Asp Pro Ile Ser Gly His
35 40 45
Val Ser Leu Phe Trp Tyr Gln Gln Ala Leu Gly Gln Gly Pro Glu Phe
50 55 60
Leu Thr Tyr Phe Gln Asn Glu Ala Gln Leu Asp Lys Ser Gly Leu Pro
65 70 75 80
Ser Asp Arg Phe Phe Ala Glu Arg Pro Glu Gly Ser Val Ser Thr Leu
85 90 95
Lys Ile Gln Arg Thr Gln Gln Glu Asp Ser Ala Val Tyr Leu Cys Ala
100 105 110
Ser Ser Pro Thr Gly Thr Glu Arg Glu Leu Phe Phe Gly Glu Gly Ser
115 120 125
Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser
305 310 315 320
Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Leu
325 330 335
Leu Leu Leu Val Pro Val Leu Glu Val Ile Phe Thr Leu Gly Gly Thr
340 345 350
Arg Ala Gln Ser Val Thr Gln Leu Asp Ser His Val Ser Val Ser Glu
355 360 365
Gly Thr Pro Val Leu Leu Arg Cys Asn Tyr Ser Ser Ser Tyr Ser Pro
370 375 380
Ser Leu Phe Trp Tyr Val Gln His Pro Asn Lys Gly Leu Gln Leu Leu
385 390 395 400
Leu Lys Tyr Thr Ser Ala Ala Thr Leu Val Lys Gly Ile Asn Gly Phe
405 410 415
Glu Ala Glu Phe Lys Lys Ser Glu Thr Ser Phe His Leu Thr Lys Pro
420 425 430
Ser Ala His Met Ser Asp Ala Ala Glu Tyr Phe Cys Val Val Arg Gly
435 440 445
Gly Lys Leu Ile Phe Gly Gln Gly Thr Glu Leu Ser Val Lys Pro Asn
450 455 460
Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser
465 470 475 480
Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn
485 490 495
Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr Val
500 505 510
Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp
515 520 525
Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile
530 535 540
Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val
545 550 555 560
Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln
565 570 575
Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly
580 585 590
Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600
<210> 342
<211> 606
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 14 полноразмерная последовательность, нативный
<400> 342
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Thr Phe Trp Gly Gln Arg Arg Thr Glu Ala Phe Phe Gly Gln Gly Thr
115 120 125
Arg Leu Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu
305 310 315 320
Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Glu Lys Asn
325 330 335
Pro Leu Ala Ala Pro Leu Leu Ile Leu Trp Phe His Leu Asp Cys Val
340 345 350
Ser Ser Ile Leu Asn Val Glu Gln Ser Pro Gln Ser Leu His Val Gln
355 360 365
Glu Gly Asp Ser Thr Asn Phe Thr Cys Ser Phe Pro Ser Ser Asn Phe
370 375 380
Tyr Ala Leu His Trp Tyr Arg Trp Glu Thr Ala Lys Ser Pro Glu Ala
385 390 395 400
Leu Phe Val Met Thr Leu Asn Gly Asp Glu Lys Lys Lys Gly Arg Ile
405 410 415
Ser Ala Thr Leu Asn Thr Lys Glu Gly Tyr Ser Tyr Leu Tyr Ile Lys
420 425 430
Gly Ser Gln Pro Glu Asp Ser Ala Thr Tyr Leu Cys Ala Ser Gln Thr
435 440 445
Gly Ala Asn Asn Leu Phe Phe Gly Thr Gly Thr Arg Leu Thr Val Ile
450 455 460
Pro Tyr Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser
465 470 475 480
Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln
485 490 495
Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys
500 505 510
Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val
515 520 525
Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn
530 535 540
Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys
545 550 555 560
Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn
565 570 575
Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val
580 585 590
Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600 605
<210> 343
<211> 335
<212> Белок
<213> Искусственная последовательность
<220>
<223> tEGFR
<400> 343
Arg Lys Val Cys Asn Gly Ile Gly Ile Gly Glu Phe Lys Asp Ser Leu
1 5 10 15
Ser Ile Asn Ala Thr Asn Ile Lys His Phe Lys Asn Cys Thr Ser Ile
20 25 30
Ser Gly Asp Leu His Ile Leu Pro Val Ala Phe Arg Gly Asp Ser Phe
35 40 45
Thr His Thr Pro Pro Leu Asp Pro Gln Glu Leu Asp Ile Leu Lys Thr
50 55 60
Val Lys Glu Ile Thr Gly Phe Leu Leu Ile Gln Ala Trp Pro Glu Asn
65 70 75 80
Arg Thr Asp Leu His Ala Phe Glu Asn Leu Glu Ile Ile Arg Gly Arg
85 90 95
Thr Lys Gln His Gly Gln Phe Ser Leu Ala Val Val Ser Leu Asn Ile
100 105 110
Thr Ser Leu Gly Leu Arg Ser Leu Lys Glu Ile Ser Asp Gly Asp Val
115 120 125
Ile Ile Ser Gly Asn Lys Asn Leu Cys Tyr Ala Asn Thr Ile Asn Trp
130 135 140
Lys Lys Leu Phe Gly Thr Ser Gly Gln Lys Thr Lys Ile Ile Ser Asn
145 150 155 160
Arg Gly Glu Asn Ser Cys Lys Ala Thr Gly Gln Val Cys His Ala Leu
165 170 175
Cys Ser Pro Glu Gly Cys Trp Gly Pro Glu Pro Arg Asp Cys Val Ser
180 185 190
Cys Arg Asn Val Ser Arg Gly Arg Glu Cys Val Asp Lys Cys Asn Leu
195 200 205
Leu Glu Gly Glu Pro Arg Glu Phe Val Glu Asn Ser Glu Cys Ile Gln
210 215 220
Cys His Pro Glu Cys Leu Pro Gln Ala Met Asn Ile Thr Cys Thr Gly
225 230 235 240
Arg Gly Pro Asp Asn Cys Ile Gln Cys Ala His Tyr Ile Asp Gly Pro
245 250 255
His Cys Val Lys Thr Cys Pro Ala Gly Val Met Gly Glu Asn Asn Thr
260 265 270
Leu Val Trp Lys Tyr Ala Asp Ala Gly His Val Cys His Leu Cys His
275 280 285
Pro Asn Cys Thr Tyr Gly Cys Thr Gly Pro Gly Leu Glu Gly Cys Pro
290 295 300
Thr Asn Gly Pro Lys Ile Pro Ser Ile Ala Thr Gly Met Val Gly Ala
305 310 315 320
Leu Leu Leu Leu Leu Val Val Ala Leu Gly Ile Gly Leu Phe Met
325 330 335
<210> 344
<211> 22
<212> Белок
<213> Искусственная последовательность
<220>
<223> F2A
<400> 344
Val Lys Gln Thr Leu Asn Phe Asp Leu Leu Lys Leu Ala Gly Asp Val
1 5 10 15
Glu Ser Asn Pro Gly Pro
20
<210> 345
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> P2A
<400> 345
Ala Thr Asn Phe Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn
1 5 10 15
Pro Gly Pro
<210> 346
<211> 20
<212> Белок
<213> Искусственная последовательность
<220>
<223> E2A
<400> 346
Gln Cys Thr Asn Tyr Ala Leu Leu Lys Leu Ala Gly Asp Val Glu Ser
1 5 10 15
Asn Pro Gly Pro
20
<210> 347
<211> 66
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCR 49/ TCR 51/ TCR 52 / TCR 53 / TCR 55 - P2A
<400> 347
ggaagcggcg ccacaaactt ctcactgctg aaacaggccg gcgacgtgga ggagaatcct 60
ggccca 66
<210> 348
<211> 4627
<212> ДНК
<213> Homo sapiens
<220>
<223> Human TCR альфа, константная область (TRAC)
<300>
<308> NCBI NG_001332.3
<309> 2016-08-31
<400> 348
atatccagaa ccctgaccct gccgtgtacc agctgagaga ctctaaatcc agtgacaagt 60
ctgtctgcct attcaccgat tttgattctc aaacaaatgt gtcacaaagt aaggattctg 120
atgtgtatat cacagacaaa actgtgctag acatgaggtc tatggacttc aagagcaaca 180
gtgctgtggc ctggagcaac aaatctgact ttgcatgtgc aaacgccttc aacaacagca 240
ttattccaga agacaccttc ttccccagcc caggtaaggg cagctttggt gccttcgcag 300
gctgtttcct tgcttcagga atggccaggt tctgcccaga gctctggtca atgatgtcta 360
aaactcctct gattggtggt ctcggcctta tccattgcca ccaaaaccct ctttttacta 420
agaaacagtg agccttgttc tggcagtcca gagaatgaca cgggaaaaaa gcagatgaag 480
agaaggtggc aggagagggc acgtggccca gcctcagtct ctccaactga gttcctgcct 540
gcctgccttt gctcagactg tttgcccctt actgctcttc taggcctcat tctaagcccc 600
ttctccaagt tgcctctcct tatttctccc tgtctgccaa aaaatctttc ccagctcact 660
aagtcagtct cacgcagtca ctcattaacc caccaatcac tgattgtgcc ggcacatgaa 720
tgcaccaggt gttgaagtgg aggaattaaa aagtcagatg aggggtgtgc ccagaggaag 780
caccattcta gttgggggag cccatctgtc agctgggaaa agtccaaata acttcagatt 840
ggaatgtgtt ttaactcagg gttgagaaaa cagctacctt caggacaaaa gtcagggaag 900
ggctctctga agaaatgcta cttgaagata ccagccctac caagggcagg gagaggaccc 960
tatagaggcc tgggacagga gctcaatgag aaaggagaag agcagcaggc atgagttgaa 1020
tgaaggaggc agggccgggt cacagggcct tctaggccat gagagggtag acagtattct 1080
aaggacgcca gaaagctgtt gatcggcttc aagcagggga gggacaccta atttgctttt 1140
cttttttttt tttttttttt tttttttttt tgagatggag ttttgctctt gttgcccagg 1200
ctggagtgca atggtgcatc ttggctcact gcaacctccg cctcccaggt tcaagtgatt 1260
ctcctgcctc agcctcccga gtagctgaga ttacaggcac ccgccaccat gcctggctaa 1320
ttttttgtat ttttagtaga gacagggttt cactatgttg gccaggctgg tctcgaactc 1380
ctgacctcag gtgatccacc cgcttcagcc tcccaaagtg ctgggattac aggcgtgagc 1440
caccacaccc ggcctgcttt tcttaaagat caatctgagt gctgtacgga gagtgggttg 1500
taagccaaga gtagaagcag aaagggagca gttgcagcag agagatgatg gaggcctggg 1560
cagggtggtg gcagggaggt aaccaacacc attcaggttt caaaggtaga accatgcagg 1620
gatgagaaag caaagagggg atcaaggaag gcagctggat tttggcctga gcagctgagt 1680
caatgatagt gccgtttact aagaagaaac caaggaaaaa atttggggtg cagggatcaa 1740
aactttttgg aacatatgaa agtacgtgtt tatactcttt atggcccttg tcactatgta 1800
tgcctcgctg cctccattgg actctagaat gaagccaggc aagagcaggg tctatgtgtg 1860
atggcacatg tggccagggt catgcaacat gtactttgta caaacagtgt atattgagta 1920
aatagaaatg gtgtccagga gccgaggtat cggtcctgcc agggccaggg gctctcccta 1980
gcaggtgctc atatgctgta agttccctcc agatctctcc acaaggaggc atggaaaggc 2040
tgtagttgtt cacctgccca agaactagga ggtctggggt gggagagtca gcctgctctg 2100
gatgctgaaa gaatgtctgt ttttcctttt agaaagttcc tgtgatgtca agctggtcga 2160
gaaaagcttt gaaacaggta agacaggggt ctagcctggg tttgcacagg attgcggaag 2220
tgatgaaccc gcaataaccc tgcctggatg agggagtggg aagaaattag tagatgtggg 2280
aatgaatgat gaggaatgga aacagcggtt caagacctgc ccagagctgg gtggggtctc 2340
tcctgaatcc ctctcaccat ctctgacttt ccattctaag cactttgagg atgagtttct 2400
agcttcaata gaccaaggac tctctcctag gcctctgtat tcctttcaac agctccactg 2460
tcaagagagc cagagagagc ttctgggtgg cccagctgtg aaatttctga gtcccttagg 2520
gatagcccta aacgaaccag atcatcctga ggacagccaa gaggttttgc cttctttcaa 2580
gacaagcaac agtactcaca taggctgtgg gcaatggtcc tgtctctcaa gaatcccctg 2640
ccactcctca cacccaccct gggcccatat tcatttccat ttgagttgtt cttattgagt 2700
catccttcct gtggtagcgg aactcactaa ggggcccatc tggacccgag gtattgtgat 2760
gataaattct gagcacctac cccatcccca gaagggctca gaaataaaat aagagccaag 2820
tctagtcggt gtttcctgtc ttgaaacaca atactgttgg ccctggaaga atgcacagaa 2880
tctgtttgta aggggatatg cacagaagct gcaagggaca ggaggtgcag gagctgcagg 2940
cctcccccac ccagcctgct ctgccttggg gaaaaccgtg ggtgtgtcct gcaggccatg 3000
caggcctggg acatgcaagc ccataaccgc tgtggcctct tggttttaca gatacgaacc 3060
taaactttca aaacctgtca gtgattgggt tccgaatcct cctcctgaaa gtggccgggt 3120
ttaatctgct catgacgctg cggctgtggt ccagctgagg tgaggggcct tgaagctggg 3180
agtggggttt agggacgcgg gtctctgggt gcatcctaag ctctgagagc aaacctccct 3240
gcagggtctt gcttttaagt ccaaagcctg agcccaccaa actctcctac ttcttcctgt 3300
tacaaattcc tcttgtgcaa taataatggc ctgaaacgct gtaaaatatc ctcatttcag 3360
ccgcctcagt tgcacttctc ccctatgagg taggaagaac agttgtttag aaacgaagaa 3420
actgaggccc cacagctaat gagtggagga agagagacac ttgtgtacac cacatgcctt 3480
gtgttgtact tctctcaccg tgtaacctcc tcatgtcctc tctccccagt acggctctct 3540
tagctcagta gaaagaagac attacactca tattacaccc caatcctggc tagagtctcc 3600
gcaccctcct cccccagggt ccccagtcgt cttgctgaca actgcatcct gttccatcac 3660
catcaaaaaa aaactccagg ctgggtgcgg gggctcacac ctgtaatccc agcactttgg 3720
gaggcagagg caggaggagc acaggagctg gagaccagcc tgggcaacac agggagaccc 3780
cgcctctaca aaaagtgaaa aaattaacca ggtgtggtgc tgcacacctg tagtcccagc 3840
tacttaagag gctgagatgg gaggatcgct tgagccctgg aatgttgagg ctacaatgag 3900
ctgtgattgc gtcactgcac tccagcctgg aagacaaagc aagatcctgt ctcaaataat 3960
aaaaaaaata agaactccag ggtacatttg ctcctagaac tctaccacat agccccaaac 4020
agagccatca ccatcacatc cctaacagtc ctgggtcttc ctcagtgtcc agcctgactt 4080
ctgttcttcc tcattccaga tctgcaagat tgtaagacag cctgtgctcc ctcgctcctt 4140
cctctgcatt gcccctcttc tccctctcca aacagaggga actctcctac ccccaaggag 4200
gtgaaagctg ctaccacctc tgtgcccccc cggcaatgcc accaactgga tcctacccga 4260
atttatgatt aagattgctg aagagctgcc aaacactgct gccaccccct ctgttccctt 4320
attgctgctt gtcactgcct gacattcacg gcagaggcaa ggctgctgca gcctcccctg 4380
gctgtgcaca ttccctcctg ctccccagag actgcctccg ccatcccaca gatgatggat 4440
cttcagtggg ttctcttggg ctctaggtcc tgcagaatgt tgtgaggggt ttattttttt 4500
ttaatagtgt tcataaagaa atacatagta ttcttcttct caagacgtgg ggggaaatta 4560
tctcattatc gaggccctgc tatgctgtgt atctgggcgt gttgtatgtc ctgctgccga 4620
tgccttc 4627
<210> 349
<211> 1448
<212> ДНК
<213> Homo sapiens
<220>
<223> Human TCR бета, константная область 1 (TRBC1)
<300>
<308> NCBI NG_001333.2
<309> 2016-08-31
<400> 349
aggacctgaa caaggtgttc ccacccgagg tcgctgtgtt tgagccatca gaagcagaga 60
tctcccacac ccaaaaggcc acactggtgt gcctggccac aggcttcttc cccgaccacg 120
tggagctgag ctggtgggtg aatgggaagg aggtgcacag tggggtcagc acagacccgc 180
agcccctcaa ggagcagccc gccctcaatg actccagata ctgcctgagc agccgcctga 240
gggtctcggc caccttctgg cagaaccccc gcaaccactt ccgctgtcaa gtccagttct 300
acgggctctc ggagaatgac gagtggaccc aggatagggc caaacccgtc acccagatcg 360
tcagcgccga ggcctggggt agagcaggtg agtggggcct ggggagatgc ctggaggaga 420
ttaggtgaga ccagctacca gggaaaatgg aaagatccag gtagcagaca agactagatc 480
caaaaagaaa ggaaccagcg cacaccatga aggagaattg ggcacctgtg gttcattctt 540
ctcccagatt ctcagcccaa cagagccaag cagctgggtc ccctttctat gtggcctgtg 600
taactctcat ctgggtggtg ccccccatcc ccctcagtgc tgccacatgc catggattgc 660
aaggacaatg tggctgacat ctgcatggca gaagaaagga ggtgctgggc tgtcagagga 720
agctggtctg ggcctgggag tctgtgccaa ctgcaaatct gactttactt ttaattgcct 780
atgaaaataa ggtctctcat ttattttcct ctccctgctt tctttcagac tgtggcttta 840
cctcgggtaa gtaagccctt ccttttcctc tccctctctc atggttcttg acctagaacc 900
aaggcatgaa gaactcacag acactggagg gtggagggtg ggagagacca gagctacctg 960
tgcacaggta cccacctgtc cttcctccgt gccaacagtg tcctaccagc aaggggtcct 1020
gtctgccacc atcctctatg agatcctgct agggaaggcc accctgtatg ctgtgctggt 1080
cagcgccctt gtgttgatgg ccatggtaag caggagggca ggatggggcc agcaggctgg 1140
aggtgacaca ctgacaccaa gcacccagaa gtatagagtc cctgccagga ttggagctgg 1200
gcagtaggga gggaagagat ttcattcagg tgcctcagaa gataacttgc acctctgtag 1260
gatcacagtg gaagggtcat gctgggaagg agaagctgga gtcaccagaa aacccaatgg 1320
atgttgtgat gagccttact atttgtgtgg tcaatgggcc ctactacttt ctctcaatcc 1380
tcacaactcc tggctcttaa taacccccaa aactttctct tctgcaggtc aagagaaagg 1440
atttctga 1448
<210> 350
<211> 610
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 15 Полноразмерная последовательность, цистеин-модифицированная
<400> 350
Met Asp Ser Trp Thr Phe Cys Cys Val Ser Leu Cys Ile Leu Val Ala
1 5 10 15
Lys His Thr Asp Ala Gly Val Ile Gln Ser Pro Arg His Glu Val Thr
20 25 30
Glu Met Gly Gln Glu Val Thr Leu Arg Cys Lys Pro Ile Ser Gly His
35 40 45
Asn Ser Leu Phe Trp Tyr Arg Gln Thr Met Met Arg Gly Leu Glu Leu
50 55 60
Leu Ile Tyr Phe Asn Asn Asn Val Pro Ile Asp Asp Ser Gly Met Pro
65 70 75 80
Glu Asp Arg Phe Ser Ala Lys Met Pro Asn Ala Ser Phe Ser Thr Leu
85 90 95
Lys Ile Gln Pro Ser Glu Pro Arg Asp Ser Ala Val Tyr Phe Cys Ala
100 105 110
Ser Ser Leu Val Gly Arg Ser Arg Thr Glu Gln Tyr Phe Gly Pro Gly
115 120 125
Thr Arg Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu
130 135 140
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
145 150 155 160
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu
165 170 175
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr
180 185 190
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
195 200 205
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
210 215 220
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
225 230 235 240
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
245 250 255
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr
260 265 270
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
275 280 285
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
290 295 300
Met Val Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe
305 310 315 320
Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met
325 330 335
Ser Leu Ser Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu Gly
340 345 350
Pro Gly Ile Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val
355 360 365
Gln Glu Lys Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp
370 375 380
Gln Ser Tyr Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met
385 390 395 400
Ile Phe Leu Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu
405 410 415
Gly Arg Tyr Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu
420 425 430
Val Ile Ser Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala
435 440 445
Met Lys Pro Gly Gly Tyr Asn Lys Leu Ile Phe Gly Ala Gly Thr Arg
450 455 460
Leu Ala Val His Pro Tyr Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln
465 470 475 480
Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp
485 490 495
Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr
500 505 510
Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser
515 520 525
Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn
530 535 540
Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro
545 550 555 560
Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp
565 570 575
Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu
580 585 590
Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp
595 600 605
Ser Ser
610
<210> 351
<211> 604
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 16 Полноразмерная последовательность, цистеин-модифицированная
<400> 351
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Ser Leu Trp Gly Arg Ser Asn Gln Pro Gln His Phe Gly Asp Gly Thr
115 120 125
Arg Leu Ser Ile Leu Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu
305 310 315 320
Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Glu Thr Leu
325 330 335
Leu Gly Leu Leu Ile Leu Trp Leu Gln Leu Gln Trp Val Ser Ser Lys
340 345 350
Gln Glu Val Thr Gln Ile Pro Ala Ala Leu Ser Val Pro Glu Gly Glu
355 360 365
Asn Leu Val Leu Asn Cys Ser Phe Thr Asp Ser Ala Ile Tyr Asn Leu
370 375 380
Gln Trp Phe Arg Gln Asp Pro Gly Lys Gly Leu Thr Ser Leu Leu Leu
385 390 395 400
Ile Gln Ser Ser Gln Arg Glu Gln Thr Ser Gly Arg Leu Asn Ala Ser
405 410 415
Leu Asp Lys Ser Ser Gly Arg Ser Thr Leu Tyr Ile Ala Ala Ser Gln
420 425 430
Pro Gly Asp Ser Ala Thr Tyr Leu Cys Ala Val Arg Pro Ala Asn Asn
435 440 445
Asn Asp Met Arg Phe Gly Ala Gly Thr Arg Leu Thr Val Lys Pro Asn
450 455 460
Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser
465 470 475 480
Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn
485 490 495
Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys Val
500 505 510
Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp
515 520 525
Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile
530 535 540
Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val
545 550 555 560
Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln
565 570 575
Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly
580 585 590
Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600
<210> 352
<211> 607
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 17 Полноразмерная последовательность, цистеин-модифицированная
<400> 352
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Ser Leu Trp Gly Arg Ser Asn Gln Pro Gln His Phe Gly Asp Gly Thr
115 120 125
Arg Leu Ser Ile Leu Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu
305 310 315 320
Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Ser Leu Ser
325 330 335
Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu Gly Pro Gly Ile
340 345 350
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
355 360 365
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Pro Ser Tyr
370 375 380
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
385 390 395 400
Ile Tyr Gln Gly Ser Tyr Asp Gln Gln Asn Ala Thr Glu Gly Arg Tyr
405 410 415
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
420 425 430
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Arg Glu
435 440 445
Gly Arg Gly Asp Lys Ile Ile Phe Gly Lys Gly Thr Arg Leu His Ile
450 455 460
Leu Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp
465 470 475 480
Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser
485 490 495
Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp
500 505 510
Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala
515 520 525
Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn
530 535 540
Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser
545 550 555 560
Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu
565 570 575
Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys
580 585 590
Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600 605
<210> 353
<211> 607
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 18 Полноразмерная последовательность, цистеин-модифицированная
<400> 353
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Ser Phe Trp Gly Arg Ser Asn Ser Pro Leu His Phe Gly Asn Gly Thr
115 120 125
Arg Leu Thr Val Thr Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu
305 310 315 320
Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Glu Thr Leu
325 330 335
Leu Gly Leu Leu Ile Leu Trp Leu Gln Leu Gln Trp Val Ser Ser Lys
340 345 350
Gln Glu Val Thr Gln Ile Pro Ala Ala Leu Ser Val Pro Glu Gly Glu
355 360 365
Asn Leu Val Leu Asn Cys Ser Phe Thr Asp Ser Ala Ile Tyr Asn Leu
370 375 380
Gln Trp Phe Arg Gln Asp Pro Gly Lys Gly Leu Thr Ser Leu Leu Leu
385 390 395 400
Ile Gln Ser Ser Gln Arg Glu Gln Thr Ser Gly Arg Leu Asn Ala Ser
405 410 415
Leu Asp Lys Ser Ser Gly Arg Ser Thr Leu Tyr Ile Ala Ala Ser Gln
420 425 430
Pro Gly Asp Ser Ala Thr Tyr Leu Cys Ala Glu Gly Asn Ala Gly Gly
435 440 445
Thr Ser Tyr Gly Lys Leu Thr Phe Gly Gln Gly Thr Ile Leu Thr Val
450 455 460
His Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp
465 470 475 480
Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser
485 490 495
Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp
500 505 510
Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala
515 520 525
Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn
530 535 540
Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser
545 550 555 560
Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu
565 570 575
Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys
580 585 590
Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600 605
<210> 354
<211> 611
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 19 Полноразмерная последовательность, цистеин-модифицированная
<400> 354
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Ser Ser Trp Gly Gln Ser Thr Gly Glu Gln Tyr Phe Gly Pro Gly Thr
115 120 125
Arg Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser
305 310 315 320
Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Ser
325 330 335
Leu Ser Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu Gly Pro
340 345 350
Gly Ile Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln
355 360 365
Glu Lys Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln
370 375 380
Ser Tyr Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile
385 390 395 400
Phe Leu Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly
405 410 415
Arg Tyr Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val
420 425 430
Ile Ser Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met
435 440 445
Arg Glu Asn Thr Gly Thr Ala Ser Lys Leu Thr Phe Gly Thr Gly Thr
450 455 460
Arg Leu Gln Val Thr Leu Asp Ile Gln Asn Pro Asp Pro Ala Val Tyr
465 470 475 480
Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr
485 490 495
Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val
500 505 510
Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys
515 520 525
Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala
530 535 540
Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser
545 550 555 560
Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr
565 570 575
Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile
580 585 590
Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu
595 600 605
Trp Ser Ser
610
<210> 355
<211> 608
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 20 Полноразмерная последовательность, цистеин-модифицированная
<400> 355
Met Leu Leu Leu Leu Leu Leu Leu Gly Pro Gly Ser Gly Leu Gly Ala
1 5 10 15
Val Val Ser Gln His Pro Ser Arg Val Ile Cys Lys Ser Gly Thr Ser
20 25 30
Val Lys Ile Glu Cys Arg Ser Leu Asp Phe Gln Ala Thr Thr Met Phe
35 40 45
Trp Tyr Arg Gln Phe Pro Lys Gln Ser Leu Met Leu Met Ala Thr Ser
50 55 60
Asn Glu Gly Ser Lys Ala Thr Tyr Glu Gln Gly Val Glu Lys Asp Lys
65 70 75 80
Phe Leu Ile Asn His Ala Ser Leu Thr Leu Ser Thr Leu Thr Val Thr
85 90 95
Ser Ala His Pro Glu Asp Ser Ser Phe Tyr Ile Cys Ser Ala Ser Ser
100 105 110
Leu Ala Arg Arg Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu
115 120 125
Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val
130 135 140
Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu
145 150 155 160
Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp
165 170 175
Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln
180 185 190
Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser
195 200 205
Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His
210 215 220
Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp
225 230 235 240
Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala
245 250 255
Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly
260 265 270
Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr
275 280 285
Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys
290 295 300
Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu
305 310 315 320
Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Asn Met Leu
325 330 335
Thr Ala Ser Leu Leu Arg Ala Val Ile Ala Ser Ile Cys Val Val Ser
340 345 350
Ser Met Ala Gln Lys Val Thr Gln Ala Gln Thr Glu Ile Ser Val Val
355 360 365
Glu Lys Glu Asp Val Thr Leu Asp Cys Val Tyr Glu Thr Arg Asp Thr
370 375 380
Thr Tyr Tyr Leu Phe Trp Tyr Lys Gln Pro Pro Ser Gly Glu Leu Val
385 390 395 400
Phe Leu Ile Arg Arg Asn Ser Phe Asp Glu Gln Asn Glu Ile Ser Gly
405 410 415
Arg Tyr Ser Trp Asn Phe Gln Lys Ser Thr Ser Ser Phe Asn Phe Thr
420 425 430
Ile Thr Ala Ser Gln Val Val Asp Ser Ala Val Tyr Phe Cys Ala Leu
435 440 445
Trp Thr Gly Ala Asn Asn Leu Phe Phe Gly Thr Gly Thr Arg Leu Thr
450 455 460
Val Ile Pro Tyr Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg
465 470 475 480
Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp
485 490 495
Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr
500 505 510
Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser
515 520 525
Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe
530 535 540
Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser
545 550 555 560
Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn
565 570 575
Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu
580 585 590
Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600 605
<210> 356
<211> 625
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 21 Полноразмерная последовательность, цистеин-модифицированная
<400> 356
Met His Arg Pro Arg Arg Pro Leu His Pro Val Ala Pro Ala Met Ser
1 5 10 15
Ile Gly Leu Leu Cys Cys Val Ala Phe Ser Leu Leu Trp Ala Ser Pro
20 25 30
Val Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Gln Val Leu Lys Thr
35 40 45
Gly Gln Ser Met Thr Leu Gln Cys Ala Gln Asp Met Asn His Asn Ser
50 55 60
Met Tyr Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu Ile Tyr
65 70 75 80
Tyr Ser Ala Ser Glu Gly Thr Thr Asp Lys Gly Glu Val Pro Asn Gly
85 90 95
Tyr Asn Val Ser Arg Leu Asn Lys Arg Glu Phe Ser Leu Arg Leu Glu
100 105 110
Ser Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser Arg Pro
115 120 125
Trp Gly Asn Gln Asn Thr Glu Ala Phe Phe Gly Gln Gly Thr Arg Leu
130 135 140
Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val
145 150 155 160
Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu
165 170 175
Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp
180 185 190
Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln
195 200 205
Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser
210 215 220
Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His
225 230 235 240
Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp
245 250 255
Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala
260 265 270
Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly
275 280 285
Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr
290 295 300
Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys
305 310 315 320
Arg Lys Asp Phe Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu Lys Gln
325 330 335
Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Ser Leu Ser Ser Leu
340 345 350
Leu Lys Val Val Thr Ala Ser Leu Trp Leu Gly Pro Gly Ile Ala Gln
355 360 365
Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys Glu Ala
370 375 380
Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln Ser Tyr Gly Leu
385 390 395 400
Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu Ile Tyr
405 410 415
Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly Arg Tyr Ser Leu
420 425 430
Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser Ala Ser
435 440 445
Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Arg Glu Gly Arg
450 455 460
Val Thr Gly Gly Gly Asn Lys Leu Thr Phe Gly Thr Gly Thr Gln Leu
465 470 475 480
Lys Val Glu Leu Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu
485 490 495
Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe
500 505 510
Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile
515 520 525
Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn
530 535 540
Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala
545 550 555 560
Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu
565 570 575
Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr
580 585 590
Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu
595 600 605
Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser
610 615 620
Ser
625
<210> 357
<211> 617
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 22 Полноразмерная последовательность, цистеин-модифицированная
<400> 357
Met Gly Pro Gly Leu Leu Cys Trp Val Leu Leu Cys Leu Leu Gly Ala
1 5 10 15
Gly Pro Val Asp Ala Gly Val Thr Gln Ser Pro Thr His Leu Ile Lys
20 25 30
Thr Arg Gly Gln His Val Thr Leu Arg Cys Ser Pro Ile Ser Gly His
35 40 45
Lys Ser Val Ser Trp Tyr Gln Gln Val Leu Gly Gln Gly Pro Gln Phe
50 55 60
Ile Phe Gln Tyr Tyr Glu Lys Glu Glu Arg Gly Arg Gly Asn Phe Pro
65 70 75 80
Asp Arg Phe Ser Ala Arg Gln Phe Pro Asn Tyr Ser Ser Glu Leu Asn
85 90 95
Val Asn Ala Leu Leu Leu Gly Asp Ser Ala Leu Tyr Leu Cys Ala Ser
100 105 110
Ser Arg Thr Glu Asn Tyr Gly Tyr Thr Phe Gly Ser Gly Thr Arg Leu
115 120 125
Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val
130 135 140
Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu
145 150 155 160
Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp
165 170 175
Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln
180 185 190
Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser
195 200 205
Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His
210 215 220
Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp
225 230 235 240
Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala
245 250 255
Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly
260 265 270
Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr
275 280 285
Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys
290 295 300
Arg Lys Asp Phe Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu Lys Gln
305 310 315 320
Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Ala Gln Glu Leu Gly
325 330 335
Met Gln Cys Gln Ala Arg Gly Ile Leu Gln Gln Met Trp Gly Val Phe
340 345 350
Leu Leu Tyr Val Ser Met Lys Met Gly Gly Thr Thr Gly Gln Asn Ile
355 360 365
Asp Gln Pro Thr Glu Met Thr Ala Thr Glu Gly Ala Ile Val Gln Ile
370 375 380
Asn Cys Thr Tyr Gln Thr Ser Gly Phe Asn Gly Leu Phe Trp Tyr Gln
385 390 395 400
Gln His Ala Gly Glu Ala Pro Thr Phe Leu Ser Tyr Asn Val Leu Asp
405 410 415
Gly Leu Glu Glu Lys Gly Arg Phe Ser Ser Phe Leu Ser Arg Ser Lys
420 425 430
Gly Tyr Ser Tyr Leu Leu Leu Lys Glu Leu Gln Met Lys Asp Ser Ala
435 440 445
Ser Tyr Leu Cys Ala Val Arg Ala Arg Met Asp Ser Asn Tyr Gln Leu
450 455 460
Ile Trp Gly Ala Gly Thr Lys Leu Ile Ile Lys Pro Asp Ile Gln Asn
465 470 475 480
Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys
485 490 495
Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln
500 505 510
Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys Val Leu Asp Met
515 520 525
Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys
530 535 540
Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu
545 550 555 560
Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val
565 570 575
Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser
580 585 590
Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu
595 600 605
Leu Met Thr Leu Arg Leu Trp Ser Ser
610 615
<210> 358
<211> 609
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 23 Полноразмерная последовательность, цистеин-модифицированная
<400> 358
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Ser Pro Trp Gly Gln Ser Asn Gln Pro Gln His Phe Gly Asp Gly Thr
115 120 125
Arg Leu Ser Ile Leu Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu
305 310 315 320
Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Ser Leu Ser
325 330 335
Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu Gly Pro Gly Ile
340 345 350
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
355 360 365
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln Ser Tyr
370 375 380
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
385 390 395 400
Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly Arg Tyr
405 410 415
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
420 425 430
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Ser Pro
435 440 445
Pro Gly Gly Ser Ala Arg Gln Leu Thr Phe Gly Ser Gly Thr Gln Leu
450 455 460
Thr Val Leu Pro Asp Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu
465 470 475 480
Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe
485 490 495
Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile
500 505 510
Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn
515 520 525
Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala
530 535 540
Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu
545 550 555 560
Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr
565 570 575
Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu
580 585 590
Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser
595 600 605
Ser
<210> 359
<211> 612
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 24 Полноразмерная последовательность, цистеин-модифицированная
<400> 359
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Ser Pro Phe Gly Arg Gly Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr
115 120 125
Arg Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser
305 310 315 320
Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Ser
325 330 335
Leu Ser Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu Gly Pro
340 345 350
Gly Ile Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln
355 360 365
Glu Lys Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln
370 375 380
Ser Tyr Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile
385 390 395 400
Phe Leu Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly
405 410 415
Arg Tyr Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val
420 425 430
Ile Ser Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met
435 440 445
Arg Glu Gly Arg Gly Asp Ser Trp Gly Lys Leu Gln Phe Gly Ala Gly
450 455 460
Thr Gln Val Val Val Thr Pro Asp Ile Gln Asn Pro Asp Pro Ala Val
465 470 475 480
Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe
485 490 495
Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp
500 505 510
Val Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe
515 520 525
Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys
530 535 540
Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro
545 550 555 560
Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu
565 570 575
Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg
580 585 590
Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg
595 600 605
Leu Trp Ser Ser
610
<210> 360
<211> 611
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 25 Полноразмерная последовательность, цистеин-модифицированная
<400> 360
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Ser Leu Trp Gly Gln Ser Asn Gln Pro Gln His Phe Gly Asp Gly Thr
115 120 125
Arg Leu Ser Ile Leu Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu
305 310 315 320
Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Ser Leu Ser
325 330 335
Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu Gly Pro Gly Ile
340 345 350
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
355 360 365
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln Ser Tyr
370 375 380
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
385 390 395 400
Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly Arg Tyr
405 410 415
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
420 425 430
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Arg Glu
435 440 445
Gly Ser Leu Thr Gly Gly Gly Asn Lys Leu Thr Phe Gly Thr Gly Thr
450 455 460
Gln Leu Lys Val Glu Leu Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr
465 470 475 480
Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr
485 490 495
Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val
500 505 510
Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys
515 520 525
Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala
530 535 540
Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser
545 550 555 560
Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr
565 570 575
Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile
580 585 590
Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu
595 600 605
Trp Ser Ser
610
<210> 361
<211> 610
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 26 Полноразмерная последовательность, цистеин-модифицированная
<400> 361
Met Ser Leu Gly Leu Leu Cys Cys Gly Ala Phe Ser Leu Leu Trp Ala
1 5 10 15
Gly Pro Val Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Arg Val Leu
20 25 30
Lys Thr Gly Gln Ser Met Thr Leu Leu Cys Ala Gln Asp Met Asn His
35 40 45
Glu Tyr Met Tyr Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu
50 55 60
Ile His Tyr Ser Val Gly Glu Gly Thr Thr Ala Lys Gly Glu Val Pro
65 70 75 80
Asp Gly Tyr Asn Val Ser Arg Leu Lys Lys Gln Asn Phe Leu Leu Gly
85 90 95
Leu Glu Ser Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser
100 105 110
Ser Tyr Tyr Ala Ser Gly Arg Asn Tyr Glu Gln Tyr Phe Gly Pro Gly
115 120 125
Thr Arg Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu
130 135 140
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
145 150 155 160
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu
165 170 175
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr
180 185 190
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
195 200 205
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
210 215 220
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
225 230 235 240
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
245 250 255
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr
260 265 270
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
275 280 285
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
290 295 300
Met Val Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe
305 310 315 320
Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met
325 330 335
Ser Leu Ser Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu Gly
340 345 350
Pro Gly Ile Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val
355 360 365
Gln Glu Lys Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp
370 375 380
Gln Ser Tyr Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met
385 390 395 400
Ile Phe Leu Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu
405 410 415
Gly Arg Tyr Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu
420 425 430
Val Ile Ser Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala
435 440 445
Met Arg Asp Ala Arg Asn Asn Asp Met Arg Phe Gly Ala Gly Thr Arg
450 455 460
Leu Thr Val Lys Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln
465 470 475 480
Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp
485 490 495
Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr
500 505 510
Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser
515 520 525
Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn
530 535 540
Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro
545 550 555 560
Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp
565 570 575
Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu
580 585 590
Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp
595 600 605
Ser Ser
610
<210> 362
<211> 618
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 27 Полноразмерная последовательность, цистеин-модифицированная
<400> 362
Met His Arg Pro Arg Arg Pro Leu His Pro Val Ala Pro Ala Met Ser
1 5 10 15
Ile Gly Leu Leu Cys Cys Val Ala Phe Ser Leu Leu Trp Ala Ser Pro
20 25 30
Val Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Gln Val Leu Lys Thr
35 40 45
Gly Gln Ser Met Thr Leu Gln Cys Ala Gln Asp Met Asn His Asn Ser
50 55 60
Met Tyr Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu Ile Tyr
65 70 75 80
Tyr Ser Ala Ser Glu Gly Thr Thr Asp Lys Gly Glu Val Pro Asn Gly
85 90 95
Tyr Asn Val Ser Arg Leu Asn Lys Arg Glu Phe Ser Leu Arg Leu Glu
100 105 110
Ser Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser Ser Glu
115 120 125
Phe Gly Ser Leu Asn Glu Lys Leu Phe Phe Gly Ser Gly Thr Gln Leu
130 135 140
Ser Val Leu Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val
145 150 155 160
Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu
165 170 175
Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp
180 185 190
Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln
195 200 205
Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser
210 215 220
Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His
225 230 235 240
Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp
245 250 255
Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala
260 265 270
Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly
275 280 285
Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr
290 295 300
Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys
305 310 315 320
Arg Lys Asp Phe Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu Lys Gln
325 330 335
Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Lys Leu Val Thr Ser
340 345 350
Ile Thr Val Leu Leu Ser Leu Gly Ile Met Gly Asp Ala Lys Thr Thr
355 360 365
Gln Pro Asn Ser Met Glu Ser Asn Glu Glu Glu Pro Val His Leu Pro
370 375 380
Cys Asn His Ser Thr Ile Ser Gly Thr Asp Tyr Ile His Trp Tyr Arg
385 390 395 400
Gln Leu Pro Ser Gln Gly Pro Glu Tyr Val Ile His Gly Leu Thr Ser
405 410 415
Asn Val Asn Asn Arg Met Ala Ser Leu Ala Ile Ala Glu Asp Arg Lys
420 425 430
Ser Ser Thr Leu Ile Leu His Arg Ala Thr Leu Arg Asp Ala Ala Val
435 440 445
Tyr Tyr Cys Ile Leu Arg Val Pro Pro Gln Ser Gly Gly Tyr Gln Lys
450 455 460
Val Thr Phe Gly Thr Gly Thr Lys Leu Gln Val Ile Pro Asn Ile Gln
465 470 475 480
Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp
485 490 495
Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser
500 505 510
Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys Val Leu Asp
515 520 525
Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn
530 535 540
Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro
545 550 555 560
Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu
565 570 575
Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu
580 585 590
Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn
595 600 605
Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
610 615
<210> 363
<211> 608
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 28 Полноразмерная последовательность, цистеин-модифицированная
<400> 363
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Ser Leu Trp Gly Arg Ser Ser Gly Asn Thr Ile Tyr Phe Gly Glu Gly
115 120 125
Ser Trp Leu Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu
130 135 140
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
145 150 155 160
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu
165 170 175
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr
180 185 190
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
195 200 205
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
210 215 220
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
225 230 235 240
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
245 250 255
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr
260 265 270
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
275 280 285
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
290 295 300
Met Val Lys Arg Lys Asp Phe Gly Ser Gly Ala Thr Asn Phe Ser Leu
305 310 315 320
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Met Ile
325 330 335
Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu Ser Trp Val
340 345 350
Trp Ser Gln Arg Lys Glu Val Glu Gln Asp Pro Gly Pro Phe Asn Val
355 360 365
Pro Glu Gly Ala Thr Val Ala Phe Asn Cys Thr Tyr Ser Asn Ser Ala
370 375 380
Ser Gln Ser Phe Phe Trp Tyr Arg Gln Asp Cys Arg Lys Glu Pro Lys
385 390 395 400
Leu Leu Met Ser Val Tyr Ser Ser Gly Asn Glu Asp Gly Arg Phe Thr
405 410 415
Ala Gln Leu Asn Arg Ala Ser Gln Tyr Ile Ser Leu Leu Ile Arg Asp
420 425 430
Ser Lys Leu Ser Asp Ser Ala Thr Tyr Leu Cys Val Val Arg Gly Gly
435 440 445
Gly Thr Ser Tyr Gly Lys Leu Thr Phe Gly Gln Gly Thr Ile Leu Thr
450 455 460
Val His Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg
465 470 475 480
Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp
485 490 495
Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr
500 505 510
Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser
515 520 525
Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe
530 535 540
Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser
545 550 555 560
Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn
565 570 575
Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu
580 585 590
Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600 605
<210> 364
<211> 611
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 29 Полноразмерная последовательность, цистеин-модифицированная
<400> 364
Met Ser Asn Gln Val Leu Cys Cys Val Val Leu Cys Leu Leu Gly Ala
1 5 10 15
Asn Thr Val Asp Gly Gly Ile Thr Gln Ser Pro Lys Tyr Leu Phe Arg
20 25 30
Lys Glu Gly Gln Asn Val Thr Leu Ser Cys Glu Gln Asn Leu Asn His
35 40 45
Asp Ala Met Tyr Trp Tyr Arg Gln Asp Pro Gly Gln Gly Leu Arg Leu
50 55 60
Ile Tyr Tyr Ser Gln Ile Val Asn Asp Phe Gln Lys Gly Asp Ile Ala
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Ser Phe Pro Leu Thr
85 90 95
Val Thr Ser Ala Gln Lys Asn Pro Thr Ala Phe Tyr Leu Cys Ala Ser
100 105 110
Ser Pro Trp Gly Arg Ala Thr Asn Glu Gln Phe Phe Gly Pro Gly Thr
115 120 125
Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser
305 310 315 320
Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Ser
325 330 335
Leu Ser Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu Gly Pro
340 345 350
Gly Ile Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln
355 360 365
Glu Lys Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln
370 375 380
Ser Tyr Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile
385 390 395 400
Phe Leu Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly
405 410 415
Arg Tyr Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val
420 425 430
Ile Ser Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met
435 440 445
Arg Leu Asn Thr Gly Thr Ala Ser Lys Leu Thr Phe Gly Thr Gly Thr
450 455 460
Arg Leu Gln Val Thr Leu Asp Ile Gln Asn Pro Asp Pro Ala Val Tyr
465 470 475 480
Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr
485 490 495
Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val
500 505 510
Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys
515 520 525
Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala
530 535 540
Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser
545 550 555 560
Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr
565 570 575
Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile
580 585 590
Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu
595 600 605
Trp Ser Ser
610
<210> 365
<211> 605
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 30 Полноразмерная последовательность, цистеин-модифицированная
<400> 365
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ser Ser Arg Gln Pro Ser Ser Gly Asn Thr Ile Tyr Phe Gly Glu Gly
115 120 125
Ser Trp Leu Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu
130 135 140
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
145 150 155 160
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu
165 170 175
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr
180 185 190
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
195 200 205
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
210 215 220
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
225 230 235 240
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
245 250 255
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr
260 265 270
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
275 280 285
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
290 295 300
Met Val Lys Arg Lys Asp Phe Gly Ser Gly Ala Thr Asn Phe Ser Leu
305 310 315 320
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Arg Leu
325 330 335
Val Ala Arg Val Thr Val Phe Leu Thr Phe Gly Thr Ile Ile Asp Ala
340 345 350
Lys Thr Thr Gln Pro Pro Ser Met Asp Cys Ala Glu Gly Arg Ala Ala
355 360 365
Asn Leu Pro Cys Asn His Ser Thr Ile Ser Gly Asn Glu Tyr Val Tyr
370 375 380
Trp Tyr Arg Gln Ile His Ser Gln Gly Pro Gln Tyr Ile Ile His Gly
385 390 395 400
Leu Lys Asn Asn Glu Thr Asn Glu Met Ala Ser Leu Ile Ile Thr Glu
405 410 415
Asp Arg Lys Ser Ser Thr Leu Ile Leu Pro His Ala Thr Leu Arg Asp
420 425 430
Thr Ala Val Tyr Tyr Cys Ile Val Arg Gly Thr Ser Val Leu Gln Gly
435 440 445
Asn Glu Lys Leu Thr Phe Gly Thr Gly Thr Arg Leu Thr Ile Ile Pro
450 455 460
Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys
465 470 475 480
Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr
485 490 495
Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys
500 505 510
Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala
515 520 525
Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser
530 535 540
Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp
545 550 555 560
Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe
565 570 575
Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala
580 585 590
Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600 605
<210> 366
<211> 614
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 31 Полноразмерная последовательность, цистеин-модифицированная
<400> 366
Met Gly Thr Arg Leu Leu Phe Trp Val Ala Phe Cys Leu Leu Gly Ala
1 5 10 15
Asp His Thr Gly Ala Gly Val Ser Gln Ser Pro Ser Asn Lys Val Thr
20 25 30
Glu Lys Gly Lys Asp Val Glu Leu Arg Cys Asp Pro Ile Ser Gly His
35 40 45
Thr Ala Leu Tyr Trp Tyr Arg Gln Ser Leu Gly Gln Gly Leu Glu Phe
50 55 60
Leu Ile Tyr Phe Gln Gly Asn Ser Ala Pro Asp Lys Ser Gly Leu Pro
65 70 75 80
Ser Asp Arg Phe Ser Ala Glu Arg Thr Gly Gly Ser Val Ser Thr Leu
85 90 95
Thr Ile Gln Arg Thr Gln Gln Glu Asp Ser Ala Val Tyr Leu Cys Ala
100 105 110
Ser Ser Arg Phe Leu Gly Ser Thr Asp Thr Gln Tyr Phe Gly Pro Gly
115 120 125
Thr Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu
130 135 140
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
145 150 155 160
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu
165 170 175
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr
180 185 190
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
195 200 205
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
210 215 220
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
225 230 235 240
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
245 250 255
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr
260 265 270
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
275 280 285
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
290 295 300
Met Val Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe
305 310 315 320
Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met
325 330 335
Ala Met Leu Leu Gly Ala Ser Val Leu Ile Leu Trp Leu Gln Pro Asp
340 345 350
Trp Val Asn Ser Gln Gln Lys Asn Asp Asp Gln Gln Val Lys Gln Asn
355 360 365
Ser Pro Ser Leu Ser Val Gln Glu Gly Arg Ile Ser Ile Leu Asn Cys
370 375 380
Asp Tyr Thr Asn Ser Met Phe Asp Tyr Phe Leu Trp Tyr Lys Lys Tyr
385 390 395 400
Pro Ala Glu Gly Pro Thr Phe Leu Ile Ser Ile Ser Ser Ile Lys Asp
405 410 415
Lys Asn Glu Asp Gly Arg Phe Thr Val Phe Leu Asn Lys Ser Ala Lys
420 425 430
His Leu Ser Leu His Ile Val Pro Ser Gln Pro Gly Asp Ser Ala Val
435 440 445
Tyr Phe Cys Ala Ala Ser Glu Arg Gly Thr Tyr Lys Tyr Ile Phe Gly
450 455 460
Thr Gly Thr Arg Leu Lys Val Leu Ala Asn Ile Gln Asn Pro Asp Pro
465 470 475 480
Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys
485 490 495
Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp
500 505 510
Ser Asp Val Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met
515 520 525
Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe
530 535 540
Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe
545 550 555 560
Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser
565 570 575
Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly
580 585 590
Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr
595 600 605
Leu Arg Leu Trp Ser Ser
610
<210> 367
<211> 602
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 32 Полноразмерная последовательность, цистеин-модифицированная
<400> 367
Met Gly Phe Arg Leu Leu Cys Cys Val Ala Phe Cys Leu Leu Gly Ala
1 5 10 15
Gly Pro Val Asp Ser Gly Val Thr Gln Thr Pro Lys His Leu Ile Thr
20 25 30
Ala Thr Gly Gln Arg Val Thr Leu Arg Cys Ser Pro Arg Ser Gly Asp
35 40 45
Leu Ser Val Tyr Trp Tyr Gln Gln Ser Leu Asp Gln Gly Leu Gln Phe
50 55 60
Leu Ile Gln Tyr Tyr Asn Gly Glu Glu Arg Ala Lys Gly Asn Ile Leu
65 70 75 80
Glu Arg Phe Ser Ala Gln Gln Phe Pro Asp Leu His Ser Glu Leu Asn
85 90 95
Leu Ser Ser Leu Glu Leu Gly Asp Ser Ala Leu Tyr Phe Cys Ala Ser
100 105 110
Ser Val Gly Gly Asp His Ser Asp Glu Gln Phe Phe Gly Pro Gly Thr
115 120 125
Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser
305 310 315 320
Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Val
325 330 335
Leu Lys Phe Ser Val Ser Ile Leu Trp Ile Gln Leu Ala Trp Val Ser
340 345 350
Thr Gln Leu Leu Glu Gln Ser Pro Gln Phe Leu Ser Ile Gln Glu Gly
355 360 365
Glu Asn Leu Thr Val Tyr Cys Asn Ser Ser Ser Val Phe Ser Ser Leu
370 375 380
Gln Trp Tyr Arg Gln Glu Pro Gly Glu Gly Pro Val Leu Leu Val Thr
385 390 395 400
Val Val Thr Gly Gly Glu Val Lys Lys Leu Lys Arg Leu Thr Phe Gln
405 410 415
Phe Gly Asp Ala Arg Lys Asp Ser Ser Leu His Ile Thr Ala Ala Gln
420 425 430
Pro Gly Asp Thr Gly Leu Tyr Leu Cys Ala Gly Gly Ser Asn Tyr Lys
435 440 445
Leu Thr Phe Gly Lys Gly Thr Leu Leu Thr Val Asn Pro Asn Ile Gln
450 455 460
Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp
465 470 475 480
Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser
485 490 495
Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys Val Leu Asp
500 505 510
Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn
515 520 525
Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro
530 535 540
Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu
545 550 555 560
Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu
565 570 575
Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn
580 585 590
Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600
<210> 368
<211> 609
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 33 Полноразмерная последовательность, цистеин-модифицированная
<400> 368
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ser Thr Pro Arg Asp Thr Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser Leu
305 310 315 320
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Met Lys
325 330 335
Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu Ser Trp Val
340 345 350
Trp Ser Gln Gln Lys Glu Val Glu Gln Asn Ser Gly Pro Leu Ser Val
355 360 365
Pro Glu Gly Ala Ile Ala Ser Leu Asn Cys Thr Tyr Ser Asp Arg Gly
370 375 380
Ser Gln Ser Phe Phe Trp Tyr Arg Gln Tyr Ser Gly Lys Ser Pro Glu
385 390 395 400
Leu Ile Met Phe Ile Tyr Ser Asn Gly Asp Lys Glu Asp Gly Arg Phe
405 410 415
Thr Ala Gln Leu Asn Lys Ala Ser Gln Tyr Val Ser Leu Leu Ile Arg
420 425 430
Asp Ser Gln Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala Val Asn Ala
435 440 445
His His Thr Gly Gly Phe Lys Thr Ile Phe Gly Ala Gly Thr Arg Leu
450 455 460
Phe Val Lys Ala Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu
465 470 475 480
Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe
485 490 495
Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile
500 505 510
Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn
515 520 525
Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala
530 535 540
Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu
545 550 555 560
Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr
565 570 575
Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu
580 585 590
Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser
595 600 605
Ser
<210> 369
<211> 601
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 34 Полноразмерная последовательность, цистеин-модифицированная
<400> 369
Met Gly Pro Gly Leu Leu Cys Trp Ala Leu Leu Cys Leu Leu Gly Ala
1 5 10 15
Gly Ser Val Glu Thr Gly Val Thr Gln Ser Pro Thr His Leu Ile Lys
20 25 30
Thr Arg Gly Gln Gln Val Thr Leu Arg Cys Ser Ser Gln Ser Gly His
35 40 45
Asn Thr Val Ser Trp Tyr Gln Gln Ala Leu Gly Gln Gly Pro Gln Phe
50 55 60
Ile Phe Gln Tyr Tyr Arg Glu Glu Glu Asn Gly Arg Gly Asn Phe Pro
65 70 75 80
Pro Arg Phe Ser Gly Leu Gln Phe Pro Asn Tyr Ser Ser Glu Leu Asn
85 90 95
Val Asn Ala Leu Glu Leu Asp Asp Ser Ala Leu Tyr Leu Cys Ala Ser
100 105 110
Ser Ser Tyr Ala Gly Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser Leu
305 310 315 320
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Lys Lys
325 330 335
Leu Leu Ala Met Ile Leu Trp Leu Gln Leu Asp Arg Leu Ser Gly Glu
340 345 350
Leu Lys Val Glu Gln Asn Pro Leu Phe Leu Ser Met Gln Glu Gly Lys
355 360 365
Asn Tyr Thr Ile Tyr Cys Asn Tyr Ser Thr Thr Ser Asp Arg Leu Tyr
370 375 380
Trp Tyr Arg Gln Asp Pro Gly Lys Ser Leu Glu Ser Leu Phe Val Leu
385 390 395 400
Leu Ser Asn Gly Ala Val Lys Gln Glu Gly Arg Leu Met Ala Ser Leu
405 410 415
Asp Thr Lys Ala Arg Leu Ser Thr Leu His Ile Thr Ala Ala Val His
420 425 430
Asp Leu Ser Ala Thr Tyr Phe Cys Ala Val Ser Gly Thr Tyr Lys Tyr
435 440 445
Ile Phe Gly Thr Gly Thr Arg Leu Lys Val Leu Ala Asn Ile Gln Asn
450 455 460
Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys
465 470 475 480
Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln
485 490 495
Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys Val Leu Asp Met
500 505 510
Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys
515 520 525
Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu
530 535 540
Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val
545 550 555 560
Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser
565 570 575
Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu
580 585 590
Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600
<210> 370
<211> 605
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 35 Полноразмерная последовательность, цистеин-модифицированная
<400> 370
Met Gly Phe Arg Leu Leu Cys Cys Val Ala Phe Cys Leu Leu Gly Ala
1 5 10 15
Gly Pro Val Asp Ser Gly Val Thr Gln Thr Pro Lys His Leu Ile Thr
20 25 30
Ala Thr Gly Gln Arg Val Thr Leu Arg Cys Ser Pro Arg Ser Gly Asp
35 40 45
Leu Ser Val Tyr Trp Tyr Gln Gln Ser Leu Asp Gln Gly Leu Gln Phe
50 55 60
Leu Ile Gln Tyr Tyr Asn Gly Glu Glu Arg Ala Lys Gly Asn Ile Leu
65 70 75 80
Glu Arg Phe Ser Ala Gln Gln Phe Pro Asp Leu His Ser Glu Leu Asn
85 90 95
Leu Ser Ser Leu Glu Leu Gly Asp Ser Ala Leu Tyr Phe Cys Ala Ser
100 105 110
Thr Thr Ser Gly Asp Ser Ser Tyr Asn Glu Gln Phe Phe Gly Pro Gly
115 120 125
Thr Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu
130 135 140
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
145 150 155 160
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu
165 170 175
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr
180 185 190
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
195 200 205
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
210 215 220
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
225 230 235 240
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
245 250 255
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr
260 265 270
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
275 280 285
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
290 295 300
Met Val Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe
305 310 315 320
Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met
325 330 335
Ala Leu Gln Ser Thr Leu Gly Ala Val Trp Leu Gly Leu Leu Leu Asn
340 345 350
Ser Leu Trp Lys Val Ala Glu Ser Lys Asp Gln Val Phe Gln Pro Ser
355 360 365
Thr Val Ala Ser Ser Glu Gly Ala Val Val Glu Ile Phe Cys Asn His
370 375 380
Ser Val Ser Asn Ala Tyr Asn Phe Phe Trp Tyr Leu His Phe Pro Gly
385 390 395 400
Cys Ala Pro Arg Leu Leu Val Lys Gly Ser Lys Pro Ser Gln Gln Gly
405 410 415
Arg Tyr Asn Met Thr Tyr Glu Arg Phe Ser Ser Ser Leu Leu Ile Leu
420 425 430
Gln Val Arg Glu Ala Asp Ala Ala Val Tyr Tyr Cys Ala Val Ala Gly
435 440 445
Asp Tyr Lys Leu Ser Phe Gly Ala Gly Thr Thr Val Thr Val Arg Ala
450 455 460
Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys
465 470 475 480
Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr
485 490 495
Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys
500 505 510
Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala
515 520 525
Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser
530 535 540
Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp
545 550 555 560
Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe
565 570 575
Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala
580 585 590
Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600 605
<210> 371
<211> 607
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 36 Полноразмерная последовательность, цистеин-модифицированная
<400> 371
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Met Thr Gly Arg Ser Asn Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser Leu
305 310 315 320
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Met Ile
325 330 335
Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu Ser Trp Val
340 345 350
Trp Ser Gln Arg Lys Glu Val Glu Gln Asp Pro Gly Pro Phe Asn Val
355 360 365
Pro Glu Gly Ala Thr Val Ala Phe Asn Cys Thr Tyr Ser Asn Ser Ala
370 375 380
Ser Gln Ser Phe Phe Trp Tyr Arg Gln Asp Cys Arg Lys Glu Pro Lys
385 390 395 400
Leu Leu Met Ser Val Tyr Ser Ser Gly Asn Glu Asp Gly Arg Phe Thr
405 410 415
Ala Gln Leu Asn Arg Ala Ser Gln Tyr Ile Ser Leu Leu Ile Arg Asp
420 425 430
Ser Lys Leu Ser Asp Ser Ala Thr Tyr Leu Cys Val Val Asn Arg Asp
435 440 445
Asn Tyr Gly Gln Asn Phe Val Phe Gly Pro Gly Thr Arg Leu Ser Val
450 455 460
Leu Pro Tyr Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp
465 470 475 480
Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser
485 490 495
Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp
500 505 510
Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala
515 520 525
Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn
530 535 540
Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser
545 550 555 560
Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu
565 570 575
Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys
580 585 590
Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600 605
<210> 372
<211> 603
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 37 Полноразмерная последовательность, цистеин-модифицированная
<400> 372
Met Gly Thr Arg Leu Leu Cys Trp Val Val Leu Gly Phe Leu Gly Thr
1 5 10 15
Asp His Thr Gly Ala Gly Val Ser Gln Ser Pro Arg Tyr Lys Val Ala
20 25 30
Lys Arg Gly Gln Asp Val Ala Leu Arg Cys Asp Pro Ile Ser Gly His
35 40 45
Val Ser Leu Phe Trp Tyr Gln Gln Ala Leu Gly Gln Gly Pro Glu Phe
50 55 60
Leu Thr Tyr Phe Gln Asn Glu Ala Gln Leu Asp Lys Ser Gly Leu Pro
65 70 75 80
Ser Asp Arg Phe Phe Ala Glu Arg Pro Glu Gly Ser Val Ser Thr Leu
85 90 95
Lys Ile Gln Arg Thr Gln Gln Glu Asp Ser Ala Val Tyr Leu Cys Ala
100 105 110
Ser Ser Leu Leu Leu Gly Ala Tyr Gly Tyr Thr Phe Gly Ser Gly Thr
115 120 125
Arg Leu Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu
305 310 315 320
Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Lys Lys Leu
325 330 335
Leu Ala Met Ile Leu Trp Leu Gln Leu Asp Arg Leu Ser Gly Glu Leu
340 345 350
Lys Val Glu Gln Asn Pro Leu Phe Leu Ser Met Gln Glu Gly Lys Asn
355 360 365
Tyr Thr Ile Tyr Cys Asn Tyr Ser Thr Thr Ser Asp Arg Leu Tyr Trp
370 375 380
Tyr Arg Gln Asp Pro Gly Lys Ser Leu Glu Ser Leu Phe Val Leu Leu
385 390 395 400
Ser Asn Gly Ala Val Lys Gln Glu Gly Arg Leu Met Ala Ser Leu Asp
405 410 415
Thr Lys Ala Arg Leu Ser Thr Leu His Ile Thr Ala Ala Val His Asp
420 425 430
Leu Ser Ala Thr Tyr Phe Cys Ala Gly Tyr Ser Gly Ala Gly Ser Tyr
435 440 445
Gln Leu Thr Phe Gly Lys Gly Thr Lys Leu Ser Val Ile Pro Asn Ile
450 455 460
Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser
465 470 475 480
Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val
485 490 495
Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys Val Leu
500 505 510
Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser
515 520 525
Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile
530 535 540
Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys
545 550 555 560
Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn
565 570 575
Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe
580 585 590
Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600
<210> 373
<211> 612
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 38 Полноразмерная последовательность, цистеин-модифицированная
<400> 373
Met Gly Pro Gly Leu Leu Cys Trp Val Leu Leu Cys Leu Leu Gly Ala
1 5 10 15
Gly Ser Val Glu Thr Gly Val Thr Gln Ser Pro Thr His Leu Ile Lys
20 25 30
Thr Arg Gly Gln Gln Val Thr Leu Arg Cys Ser Ser Gln Ser Gly His
35 40 45
Asn Thr Val Ser Trp Tyr Gln Gln Ala Leu Gly Gln Gly Pro Gln Phe
50 55 60
Ile Phe Gln Tyr Tyr Arg Glu Glu Glu Asn Gly Arg Gly Asn Phe Pro
65 70 75 80
Pro Arg Phe Ser Gly Leu Gln Phe Pro Asn Tyr Ser Ser Glu Leu Asn
85 90 95
Val Asn Ala Leu Glu Leu Asp Asp Ser Ala Leu Tyr Leu Cys Ala Ser
100 105 110
Ser Leu Val Ala Gly Gly Glu Thr Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Leu Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser Leu
305 310 315 320
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Val Lys
325 330 335
Met Pro Gly Ala Arg Arg Gln Ser Ile Met Lys Arg Ile Leu Gly Ala
340 345 350
Leu Leu Gly Leu Leu Ser Ala Gln Val Cys Cys Val Arg Gly Ile Gln
355 360 365
Val Glu Gln Ser Pro Pro Asp Leu Ile Leu Gln Glu Gly Ala Asn Ser
370 375 380
Thr Leu Arg Cys Asn Phe Ser Asp Ser Val Asn Asn Leu Gln Trp Phe
385 390 395 400
His Gln Asn Pro Trp Gly Gln Leu Ile Asn Leu Phe Tyr Ile Pro Ser
405 410 415
Gly Thr Lys Gln Asn Gly Arg Leu Ser Ala Thr Thr Val Ala Thr Glu
420 425 430
Arg Tyr Ser Leu Leu Tyr Ile Ser Ser Ser Gln Thr Thr Asp Ser Gly
435 440 445
Val Tyr Phe Cys Ala Val Gly Phe Asn Asp Met Arg Phe Gly Ala Gly
450 455 460
Thr Arg Leu Thr Val Lys Pro Asn Ile Gln Asn Pro Asp Pro Ala Val
465 470 475 480
Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe
485 490 495
Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp
500 505 510
Val Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe
515 520 525
Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys
530 535 540
Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro
545 550 555 560
Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu
565 570 575
Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg
580 585 590
Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg
595 600 605
Leu Trp Ser Ser
610
<210> 374
<211> 592
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 39 Полноразмерная последовательность, цистеин-модифицированная
<400> 374
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ser Thr Pro Arg Asp Arg Gly Lys Glu Ala Phe Phe Gly Gln Gly Thr
115 120 125
Arg Leu Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu
305 310 315 320
Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Gln Leu Thr
325 330 335
Trp Val Ser Gly Gln Gln Leu Asn Gln Ser Pro Gln Ser Met Phe Ile
340 345 350
Gln Glu Gly Glu Asp Val Ser Met Asn Cys Thr Ser Ser Ser Ile Phe
355 360 365
Asn Thr Trp Leu Trp Tyr Lys Gln Glu Pro Gly Glu Gly Pro Val Leu
370 375 380
Leu Ile Ala Leu Tyr Lys Ala Gly Glu Leu Thr Ser Asn Gly Arg Leu
385 390 395 400
Thr Ala Gln Phe Gly Ile Thr Arg Lys Asp Ser Phe Leu Asn Ile Ser
405 410 415
Ala Ser Ile Pro Ser Asp Val Gly Ile Tyr Phe Cys Ala Gly Tyr Ser
420 425 430
Ser Ser Asn Asp Tyr Lys Leu Ser Phe Gly Ala Gly Thr Thr Val Thr
435 440 445
Val Arg Ala Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg
450 455 460
Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp
465 470 475 480
Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr
485 490 495
Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser
500 505 510
Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe
515 520 525
Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser
530 535 540
Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn
545 550 555 560
Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu
565 570 575
Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
580 585 590
<210> 375
<211> 638
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 40 Полноразмерная последовательность, цистеин-модифицированная
<400> 375
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ile Thr Ala Arg Ser Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser Leu
305 310 315 320
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met His Thr
325 330 335
Ser Thr Phe Gln Asn Arg Pro Gln Leu Phe Leu Leu Ile Trp Lys Lys
340 345 350
Leu Val Pro Gly Asn Pro Phe Arg Arg Ser Trp Met Lys Arg Glu Arg
355 360 365
Glu Met Leu Leu Ile Thr Ser Met Leu Val Leu Trp Met Gln Leu Ser
370 375 380
Gln Val Asn Gly Gln Gln Val Met Gln Ile Pro Gln Tyr Gln His Val
385 390 395 400
Gln Glu Gly Glu Asp Phe Thr Thr Tyr Cys Asn Ser Ser Thr Thr Leu
405 410 415
Ser Asn Ile Gln Trp Tyr Lys Gln Arg Pro Gly Gly His Pro Val Phe
420 425 430
Leu Ile Gln Leu Val Lys Ser Gly Glu Val Lys Lys Gln Lys Arg Leu
435 440 445
Thr Phe Gln Phe Gly Glu Ala Lys Lys Asn Ser Ser Leu His Ile Thr
450 455 460
Ala Thr Gln Thr Thr Asp Val Gly Thr Tyr Phe Cys Ala Gly Arg Asn
465 470 475 480
Asn Phe Asn Lys Phe Tyr Phe Gly Ser Gly Thr Lys Leu Asn Val Lys
485 490 495
Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser
500 505 510
Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln
515 520 525
Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys
530 535 540
Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val
545 550 555 560
Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn
565 570 575
Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys
580 585 590
Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn
595 600 605
Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val
610 615 620
Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
625 630 635
<210> 376
<211> 610
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 41 Полноразмерная последовательность, цистеин-модифицированная
<400> 376
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ser Asn Pro Arg Asp Arg Val Ser Tyr Glu Gln Tyr Phe Gly Pro Gly
115 120 125
Thr Arg Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu
130 135 140
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
145 150 155 160
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu
165 170 175
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr
180 185 190
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
195 200 205
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
210 215 220
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
225 230 235 240
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
245 250 255
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr
260 265 270
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
275 280 285
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
290 295 300
Met Val Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe
305 310 315 320
Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met
325 330 335
Met Ile Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu Ser
340 345 350
Trp Val Trp Ser Gln Arg Lys Glu Val Glu Gln Asp Pro Gly Pro Phe
355 360 365
Asn Val Pro Glu Gly Ala Thr Val Ala Phe Asn Cys Thr Tyr Ser Asn
370 375 380
Ser Ala Ser Gln Ser Phe Phe Trp Tyr Arg Gln Asp Cys Arg Lys Glu
385 390 395 400
Pro Lys Leu Leu Met Ser Val Tyr Ser Ser Gly Asn Glu Asp Gly Arg
405 410 415
Phe Thr Ala Gln Leu Asn Arg Ala Ser Gln Tyr Ile Ser Leu Leu Ile
420 425 430
Arg Asp Ser Lys Leu Ser Asp Ser Ala Thr Tyr Leu Cys Val Val Thr
435 440 445
Phe Ala Leu Thr Gly Gly Phe Lys Thr Ile Phe Gly Ala Gly Thr Arg
450 455 460
Leu Phe Val Lys Ala Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln
465 470 475 480
Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp
485 490 495
Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr
500 505 510
Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser
515 520 525
Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn
530 535 540
Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro
545 550 555 560
Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp
565 570 575
Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu
580 585 590
Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp
595 600 605
Ser Ser
610
<210> 377
<211> 638
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 42 Полноразмерная последовательность, цистеин-модифицированная
<400> 377
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Lys Thr Ser Arg Ser Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser Leu
305 310 315 320
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met His Thr
325 330 335
Ser Thr Phe Gln Asn Arg Pro Gln Leu Phe Leu Leu Ile Trp Lys Lys
340 345 350
Leu Val Pro Gly Asn Pro Phe Arg Arg Ser Trp Met Lys Arg Glu Arg
355 360 365
Glu Met Leu Leu Ile Thr Ser Met Leu Val Leu Trp Met Gln Leu Ser
370 375 380
Gln Val Asn Gly Gln Gln Val Met Gln Ile Pro Gln Tyr Gln His Val
385 390 395 400
Gln Glu Gly Glu Asp Phe Thr Thr Tyr Cys Asn Ser Ser Thr Thr Leu
405 410 415
Ser Asn Ile Gln Trp Tyr Lys Gln Arg Pro Gly Gly His Pro Val Phe
420 425 430
Leu Ile Gln Leu Val Lys Ser Gly Glu Val Lys Lys Gln Lys Arg Leu
435 440 445
Thr Phe Gln Phe Gly Glu Ala Lys Lys Asn Ser Ser Leu His Ile Thr
450 455 460
Ala Thr Gln Thr Thr Asp Val Gly Thr Tyr Phe Cys Ala Gly Pro Asp
465 470 475 480
Asn Phe Asn Lys Phe Tyr Phe Gly Ser Gly Thr Lys Leu Asn Val Lys
485 490 495
Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser
500 505 510
Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln
515 520 525
Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys
530 535 540
Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val
545 550 555 560
Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn
565 570 575
Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys
580 585 590
Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn
595 600 605
Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val
610 615 620
Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
625 630 635
<210> 378
<211> 609
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 43 Полноразмерная последовательность, цистеин-модифицированная
<400> 378
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ser Thr Pro Arg Asp Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser Leu
305 310 315 320
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Met Lys
325 330 335
Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu Ser Trp Val
340 345 350
Trp Ser Gln Gln Lys Glu Val Glu Gln Asn Ser Gly Pro Leu Ser Val
355 360 365
Pro Glu Gly Ala Ile Ala Ser Leu Asn Cys Thr Tyr Ser Asp Arg Gly
370 375 380
Ser Gln Ser Phe Phe Trp Tyr Arg Gln Tyr Ser Gly Lys Ser Pro Glu
385 390 395 400
Leu Ile Met Phe Ile Tyr Ser Asn Gly Asp Lys Glu Asp Gly Arg Phe
405 410 415
Thr Ala Gln Leu Asn Lys Ala Ser Gln Tyr Val Ser Leu Leu Ile Arg
420 425 430
Asp Ser Gln Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala Val Asn Val
435 440 445
Pro Thr Ser Gly Thr Tyr Lys Tyr Ile Phe Gly Thr Gly Thr Arg Leu
450 455 460
Lys Val Leu Ala Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu
465 470 475 480
Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe
485 490 495
Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile
500 505 510
Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn
515 520 525
Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala
530 535 540
Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu
545 550 555 560
Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr
565 570 575
Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu
580 585 590
Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser
595 600 605
Ser
<210> 379
<211> 616
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 44 Полноразмерная последовательность, цистеин-модифицированная
<400> 379
Met Gly Phe Arg Leu Leu Cys Cys Val Ala Phe Cys Leu Leu Gly Ala
1 5 10 15
Gly Pro Val Asp Ser Gly Val Thr Gln Thr Pro Lys His Leu Ile Thr
20 25 30
Ala Thr Gly Gln Arg Val Thr Leu Arg Cys Ser Pro Arg Ser Gly Asp
35 40 45
Leu Ser Val Tyr Trp Tyr Gln Gln Ser Leu Asp Gln Gly Leu Gln Phe
50 55 60
Leu Ile Gln Tyr Tyr Asn Gly Glu Glu Arg Ala Lys Gly Asn Ile Leu
65 70 75 80
Glu Arg Phe Ser Ala Gln Gln Phe Pro Asp Leu His Ser Glu Leu Asn
85 90 95
Leu Ser Ser Leu Glu Leu Gly Asp Ser Ala Leu Tyr Phe Cys Ala Ser
100 105 110
Ser Gly Thr Pro Asp Thr Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr
115 120 125
Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe
130 135 140
Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val
145 150 155 160
Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp
165 170 175
Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro
180 185 190
Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser
195 200 205
Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe
210 215 220
Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr
225 230 235 240
Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp
245 250 255
Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val
260 265 270
Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu
275 280 285
Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg
290 295 300
Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu Lys
305 310 315 320
Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Ala Gln Glu Leu
325 330 335
Gly Met Gln Cys Gln Ala Arg Gly Ile Leu Gln Gln Met Trp Gly Val
340 345 350
Phe Leu Leu Tyr Val Ser Met Lys Met Gly Gly Thr Thr Gly Gln Asn
355 360 365
Ile Asp Gln Pro Thr Glu Met Thr Ala Thr Glu Gly Ala Ile Val Gln
370 375 380
Ile Asn Cys Thr Tyr Gln Thr Ser Gly Phe Asn Gly Leu Phe Trp Tyr
385 390 395 400
Gln Gln His Ala Gly Glu Ala Pro Thr Phe Leu Ser Tyr Asn Val Leu
405 410 415
Asp Gly Leu Glu Glu Lys Gly Arg Phe Ser Ser Phe Leu Ser Arg Ser
420 425 430
Lys Gly Tyr Ser Tyr Leu Leu Leu Lys Glu Leu Gln Met Lys Asp Ser
435 440 445
Ala Ser Tyr Leu Cys Ala Gln Tyr Ser Gly Gly Tyr Gln Lys Val Thr
450 455 460
Phe Gly Thr Gly Thr Lys Leu Gln Val Ile Pro Asn Ile Gln Asn Pro
465 470 475 480
Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser
485 490 495
Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser
500 505 510
Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg
515 520 525
Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser
530 535 540
Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp
545 550 555 560
Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu
565 570 575
Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val
580 585 590
Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu
595 600 605
Met Thr Leu Arg Leu Trp Ser Ser
610 615
<210> 380
<211> 642
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 45 Полноразмерная последовательность, цистеин-модифицированная
<400> 380
Met Gly Thr Arg Leu Leu Phe Trp Val Ala Phe Cys Leu Leu Gly Ala
1 5 10 15
Tyr His Thr Gly Ala Gly Val Ser Gln Ser Pro Ser Asn Lys Val Thr
20 25 30
Glu Lys Gly Lys Asp Val Glu Leu Arg Cys Asp Pro Ile Ser Gly His
35 40 45
Thr Ala Leu Tyr Trp Tyr Arg Gln Arg Leu Gly Gln Gly Leu Glu Phe
50 55 60
Leu Ile Tyr Phe Gln Gly Asn Ser Ala Pro Asp Lys Ser Gly Leu Pro
65 70 75 80
Ser Asp Arg Phe Ser Ala Glu Arg Thr Gly Glu Ser Val Ser Thr Leu
85 90 95
Thr Ile Gln Arg Thr Gln Gln Glu Asp Ser Ala Val Tyr Leu Cys Ala
100 105 110
Ser Ser Leu Tyr Leu Gly Thr Thr Gly Glu Leu Phe Phe Gly Glu Gly
115 120 125
Ser Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu
130 135 140
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
145 150 155 160
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu
165 170 175
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr
180 185 190
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
195 200 205
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
210 215 220
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
225 230 235 240
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
245 250 255
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr
260 265 270
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
275 280 285
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
290 295 300
Met Val Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe
305 310 315 320
Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met
325 330 335
His Thr Ser Thr Phe Gln Asn Arg Pro Gln Leu Phe Leu Leu Ile Trp
340 345 350
Lys Lys Leu Val Pro Gly Asn Pro Phe Arg Arg Ser Trp Met Lys Arg
355 360 365
Glu Arg Glu Met Leu Leu Ile Thr Ser Met Leu Val Leu Trp Met Gln
370 375 380
Leu Ser Gln Val Asn Gly Gln Gln Val Met Gln Ile Pro Gln Tyr Gln
385 390 395 400
His Val Gln Glu Gly Glu Asp Phe Thr Thr Tyr Cys Asn Ser Ser Thr
405 410 415
Thr Leu Ser Asn Ile Gln Trp Tyr Lys Gln Arg Pro Gly Gly His Pro
420 425 430
Val Phe Leu Ile Gln Leu Val Lys Ser Gly Glu Val Lys Lys Gln Lys
435 440 445
Arg Leu Thr Phe Gln Phe Gly Glu Ala Lys Lys Asn Ser Ser Leu His
450 455 460
Ile Thr Ala Thr Gln Thr Thr Asp Val Gly Thr Tyr Phe Cys Ala Gly
465 470 475 480
Ser Ser Gly Ala Gly Ser Tyr Gln Leu Thr Phe Gly Lys Gly Thr Lys
485 490 495
Leu Ser Val Ile Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln
500 505 510
Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp
515 520 525
Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr
530 535 540
Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser
545 550 555 560
Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn
565 570 575
Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro
580 585 590
Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp
595 600 605
Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu
610 615 620
Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp
625 630 635 640
Ser Ser
<210> 381
<211> 609
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 46 Полноразмерная последовательность, цистеин-модифицированная
<400> 381
Met Gly Thr Arg Leu Leu Cys Trp Ala Ala Leu Cys Leu Leu Gly Ala
1 5 10 15
Asp His Thr Gly Ala Gly Val Ser Gln Thr Pro Ser Asn Lys Val Thr
20 25 30
Glu Lys Gly Lys Tyr Val Glu Leu Arg Cys Asp Pro Ile Ser Gly His
35 40 45
Thr Ala Leu Tyr Trp Tyr Arg Gln Ser Leu Gly Gln Gly Pro Glu Phe
50 55 60
Leu Ile Tyr Phe Gln Gly Thr Gly Ala Ala Asp Asp Ser Gly Leu Pro
65 70 75 80
Asn Asp Arg Phe Phe Ala Val Arg Pro Glu Gly Ser Val Ser Thr Leu
85 90 95
Lys Ile Gln Arg Thr Glu Arg Gly Asp Ser Ala Val Tyr Leu Cys Ala
100 105 110
Ser Ser Leu Tyr Leu Gly Gly Ser Glu Thr Gln Tyr Phe Gly Pro Gly
115 120 125
Thr Arg Leu Leu Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu
130 135 140
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
145 150 155 160
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu
165 170 175
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr
180 185 190
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
195 200 205
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
210 215 220
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
225 230 235 240
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
245 250 255
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr
260 265 270
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
275 280 285
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
290 295 300
Met Val Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe
305 310 315 320
Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met
325 330 335
Leu Leu Leu Leu Val Pro Ala Phe Gln Val Ile Phe Thr Leu Gly Gly
340 345 350
Thr Arg Ala Gln Ser Val Thr Gln Leu Asp Ser Gln Val Pro Val Phe
355 360 365
Glu Glu Ala Pro Val Glu Leu Arg Cys Asn Tyr Ser Ser Ser Val Ser
370 375 380
Val Tyr Leu Phe Trp Tyr Val Gln Tyr Pro Asn Gln Gly Leu Gln Leu
385 390 395 400
Leu Leu Lys Tyr Leu Ser Gly Ser Thr Leu Val Lys Gly Ile Asn Gly
405 410 415
Phe Glu Ala Glu Phe Asn Lys Ser Gln Thr Ser Phe His Leu Arg Lys
420 425 430
Pro Ser Val His Ile Ser Asp Thr Ala Glu Tyr Phe Cys Ala Val Ser
435 440 445
Pro Ser Ser Gly Thr Tyr Lys Tyr Ile Phe Gly Thr Gly Thr Arg Leu
450 455 460
Lys Val Leu Ala Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu
465 470 475 480
Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe
485 490 495
Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile
500 505 510
Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn
515 520 525
Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala
530 535 540
Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu
545 550 555 560
Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr
565 570 575
Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu
580 585 590
Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser
595 600 605
Ser
<210> 382
<211> 609
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 47 Полноразмерная последовательность, цистеин-модифицированная
<400> 382
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Met Thr Gly Arg Thr Thr Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser Leu
305 310 315 320
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Met Lys
325 330 335
Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu Ser Trp Val
340 345 350
Trp Ser Gln Gln Lys Glu Val Glu Gln Asn Ser Gly Pro Leu Ser Val
355 360 365
Pro Glu Gly Ala Ile Ala Ser Leu Asn Cys Thr Tyr Ser Asp Arg Gly
370 375 380
Ser Gln Ser Phe Phe Trp Tyr Arg Gln Tyr Ser Gly Lys Ser Pro Glu
385 390 395 400
Leu Ile Met Phe Ile Tyr Ser Asn Gly Asp Lys Glu Asp Gly Arg Phe
405 410 415
Thr Ala Gln Leu Asn Lys Ala Ser Gln Tyr Val Ser Leu Leu Ile Arg
420 425 430
Asp Ser Gln Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala Val Asn Leu
435 440 445
Leu Ser Gly Ser Ala Arg Gln Leu Thr Phe Gly Ser Gly Thr Gln Leu
450 455 460
Thr Val Leu Pro Asp Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu
465 470 475 480
Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe
485 490 495
Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile
500 505 510
Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn
515 520 525
Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala
530 535 540
Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu
545 550 555 560
Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr
565 570 575
Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu
580 585 590
Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser
595 600 605
Ser
<210> 383
<211> 607
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 48 Полноразмерная последовательность, цистеин-модифицированная
<400> 383
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ser Thr Gly Arg Val Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser Leu
305 310 315 320
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Met Lys
325 330 335
Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu Ser Trp Val
340 345 350
Trp Ser Gln Gln Lys Glu Val Glu Gln Asp Pro Gly Pro Leu Ser Val
355 360 365
Pro Glu Gly Ala Ile Val Ser Leu Asn Cys Thr Tyr Ser Asn Ser Ala
370 375 380
Phe Gln Tyr Phe Met Trp Tyr Arg Gln Tyr Ser Arg Lys Gly Pro Glu
385 390 395 400
Leu Leu Met Tyr Thr Tyr Ser Ser Gly Asn Lys Glu Asp Gly Arg Phe
405 410 415
Thr Ala Gln Val Asp Lys Ser Ser Lys Tyr Ile Ser Leu Phe Ile Arg
420 425 430
Asp Ser Gln Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala Met Arg Ile
435 440 445
Gln Gly Ala Gln Lys Leu Val Phe Gly Gln Gly Thr Arg Leu Thr Ile
450 455 460
Asn Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp
465 470 475 480
Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser
485 490 495
Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp
500 505 510
Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala
515 520 525
Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn
530 535 540
Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser
545 550 555 560
Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu
565 570 575
Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys
580 585 590
Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600 605
<210> 384
<211> 609
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 49 Полноразмерная последовательность, цистеин-модифицированная
<400> 384
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ser Thr Pro Arg Tyr Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser Leu
305 310 315 320
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Met Lys
325 330 335
Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu Ser Trp Val
340 345 350
Trp Ser Gln Gln Lys Glu Val Glu Gln Asn Ser Gly Pro Leu Ser Val
355 360 365
Pro Glu Gly Ala Ile Ala Ser Leu Asn Cys Thr Tyr Ser Asp Arg Gly
370 375 380
Ser Gln Ser Phe Phe Trp Tyr Arg Gln Tyr Ser Gly Lys Ser Pro Glu
385 390 395 400
Leu Ile Met Phe Ile Tyr Ser Asn Gly Asp Lys Glu Asp Gly Arg Phe
405 410 415
Thr Ala Gln Leu Asn Lys Ala Ser Gln Tyr Val Ser Leu Leu Ile Arg
420 425 430
Asp Ser Gln Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala Val Asn Ile
435 440 445
Gly Thr Ser Gly Thr Tyr Lys Tyr Ile Phe Gly Thr Gly Thr Arg Leu
450 455 460
Lys Val Leu Ala Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu
465 470 475 480
Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe
485 490 495
Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile
500 505 510
Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn
515 520 525
Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala
530 535 540
Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu
545 550 555 560
Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr
565 570 575
Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu
580 585 590
Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser
595 600 605
Ser
<210> 385
<211> 604
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 50 Полноразмерная последовательность, цистеин-модифицированная
<400> 385
Met Gly Phe Arg Leu Leu Cys Cys Val Ala Phe Cys Leu Leu Gly Ala
1 5 10 15
Gly Pro Val Asp Ser Gly Val Thr Gln Thr Pro Lys His Leu Ile Thr
20 25 30
Ala Thr Gly Gln Arg Val Thr Leu Arg Cys Ser Pro Arg Ser Gly Asp
35 40 45
Leu Ser Val Tyr Trp Tyr Gln Gln Ser Leu Asp Gln Gly Leu Gln Phe
50 55 60
Leu Ile His Tyr Tyr Asn Gly Glu Glu Arg Ala Lys Gly Asn Ile Leu
65 70 75 80
Glu Arg Phe Ser Ala Gln Gln Phe Pro Asp Leu His Ser Glu Leu Asn
85 90 95
Leu Ser Ser Leu Glu Leu Gly Asp Ser Ala Leu Tyr Phe Cys Ala Ser
100 105 110
Ser Ala Thr Arg Asp Ala Tyr Gly Tyr Thr Phe Gly Ser Gly Thr Arg
115 120 125
Leu Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Phe Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu Lys
305 310 315 320
Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Lys Thr Phe Ala
325 330 335
Gly Phe Ser Phe Leu Phe Leu Trp Leu Gln Leu Asp Cys Met Ser Arg
340 345 350
Gly Glu Asp Val Glu Gln Ser Leu Phe Leu Ser Val Arg Glu Gly Asp
355 360 365
Ser Ser Val Ile Asn Cys Thr Tyr Thr Asp Ser Ser Ser Thr Tyr Leu
370 375 380
Tyr Trp Tyr Lys Gln Glu Pro Gly Ala Gly Leu Gln Leu Leu Thr Tyr
385 390 395 400
Ile Phe Ser Asn Met Asp Met Lys Gln Asp Gln Arg Leu Thr Val Leu
405 410 415
Leu Asn Lys Lys Asp Lys His Leu Ser Leu Arg Ile Ala Asp Thr Gln
420 425 430
Thr Gly Asp Ser Ala Ile Tyr Phe Cys Ala Glu Ser Pro Pro Gly Thr
435 440 445
Tyr Lys Tyr Ile Phe Gly Thr Gly Thr Arg Leu Lys Val Leu Ala Asn
450 455 460
Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser
465 470 475 480
Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn
485 490 495
Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys Val
500 505 510
Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp
515 520 525
Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile
530 535 540
Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val
545 550 555 560
Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln
565 570 575
Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly
580 585 590
Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600
<210> 386
<211> 610
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 51 Полноразмерная последовательность, цистеин-модифицированная
<400> 386
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ile Ala Ser Arg Val Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser Leu
305 310 315 320
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Met Lys
325 330 335
Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu Ser Trp Val
340 345 350
Trp Ser Gln Gln Lys Glu Val Glu Gln Asn Ser Gly Pro Leu Ser Val
355 360 365
Pro Glu Gly Ala Ile Ala Ser Leu Asn Cys Thr Tyr Ser Asp Arg Gly
370 375 380
Ser Gln Ser Phe Phe Trp Tyr Arg Gln Tyr Ser Gly Lys Ser Pro Glu
385 390 395 400
Leu Ile Met Ser Ile Tyr Ser Asn Gly Asp Lys Glu Asp Gly Arg Phe
405 410 415
Thr Ala Gln Leu Asn Lys Ala Ser Gln Tyr Val Ser Leu Leu Ile Arg
420 425 430
Asp Ser Gln Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala Val Asn Met
435 440 445
Arg Gly Gly Gly Ser Asn Tyr Lys Leu Thr Phe Gly Lys Gly Thr Leu
450 455 460
Leu Thr Val Asn Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln
465 470 475 480
Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp
485 490 495
Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr
500 505 510
Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser
515 520 525
Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn
530 535 540
Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro
545 550 555 560
Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp
565 570 575
Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu
580 585 590
Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp
595 600 605
Ser Ser
610
<210> 387
<211> 601
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 52 Полноразмерная последовательность, цистеин-модифицированная
<400> 387
Met Gly Phe Arg Leu Leu Cys Cys Val Ala Phe Cys Leu Leu Gly Ala
1 5 10 15
Gly Pro Val Asp Ser Gly Val Thr Gln Thr Pro Lys His Leu Ile Thr
20 25 30
Ala Thr Gly Gln Arg Val Thr Leu Arg Cys Ser Pro Arg Ser Gly Asp
35 40 45
Leu Ser Val Tyr Trp Tyr Gln Gln Ser Leu Asp Gln Gly Leu Gln Phe
50 55 60
Leu Ile Gln Tyr Tyr Asn Gly Glu Glu Arg Ala Lys Gly Asn Ile Leu
65 70 75 80
Glu Arg Phe Ser Ala Gln Gln Phe Pro Asp Leu His Ser Glu Leu Asn
85 90 95
Leu Ser Ser Leu Glu Leu Gly Asp Ser Ala Leu Tyr Phe Cys Ala Ser
100 105 110
Ser Val Gly Asp Leu Asn Asn Glu Gln Phe Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser Leu
305 310 315 320
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Val Leu
325 330 335
Lys Phe Ser Val Ser Ile Leu Trp Ile Gln Leu Ala Trp Val Ser Thr
340 345 350
Gln Leu Leu Glu Gln Ser Pro Gln Phe Leu Ser Ile Gln Glu Gly Glu
355 360 365
Asn Leu Thr Val Tyr Cys Asn Ser Ser Ser Val Phe Ser Ser Leu Gln
370 375 380
Trp Tyr Arg Gln Glu Pro Gly Glu Gly Pro Val Leu Leu Val Thr Val
385 390 395 400
Val Thr Gly Gly Glu Val Lys Lys Leu Lys Arg Leu Thr Phe Gln Phe
405 410 415
Gly Asp Ala Arg Lys Asp Ser Ser Leu His Ile Thr Ala Ala Gln Pro
420 425 430
Gly Asp Thr Gly Leu Tyr Leu Cys Ala Gly Ala Arg Asp Tyr Lys Leu
435 440 445
Ser Phe Gly Ala Gly Thr Thr Val Thr Val Arg Ala Asn Ile Gln Asn
450 455 460
Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys
465 470 475 480
Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln
485 490 495
Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys Val Leu Asp Met
500 505 510
Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys
515 520 525
Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu
530 535 540
Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val
545 550 555 560
Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser
565 570 575
Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu
580 585 590
Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600
<210> 388
<211> 611
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 53 Полноразмерная последовательность, цистеин-модифицированная
<400> 388
Met Gly Thr Ser Leu Leu Cys Trp Val Val Leu Gly Phe Leu Gly Thr
1 5 10 15
Asp His Thr Gly Ala Gly Val Ser Gln Ser Pro Arg Tyr Lys Val Thr
20 25 30
Lys Arg Gly Gln Asp Val Ala Leu Arg Cys Asp Pro Ile Ser Gly His
35 40 45
Val Ser Leu Tyr Trp Tyr Arg Gln Ala Leu Gly Gln Gly Pro Glu Phe
50 55 60
Leu Thr Tyr Phe Asn Tyr Glu Ala Gln Gln Asp Lys Ser Gly Leu Pro
65 70 75 80
Asn Asp Arg Phe Ser Ala Glu Arg Pro Glu Gly Ser Ile Ser Thr Leu
85 90 95
Thr Ile Gln Arg Thr Glu Gln Arg Asp Ser Ala Met Tyr Arg Cys Ala
100 105 110
Ser Ser Gly Ser Gly Thr Ser Gly Tyr Asn Glu Gln Phe Phe Gly Pro
115 120 125
Gly Thr Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro
130 135 140
Glu Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln
145 150 155 160
Lys Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val
165 170 175
Glu Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys
180 185 190
Thr Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg
195 200 205
Tyr Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn
210 215 220
Pro Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu
225 230 235 240
Asn Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val
245 250 255
Ser Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser
260 265 270
Tyr Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu
275 280 285
Gly Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met
290 295 300
Ala Met Val Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn
305 310 315 320
Phe Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro
325 330 335
Met Ala Ser Ala Pro Ile Ser Met Leu Ala Met Leu Phe Thr Leu Ser
340 345 350
Gly Leu Arg Ala Gln Ser Val Ala Gln Pro Glu Asp Gln Val Asn Val
355 360 365
Ala Glu Gly Asn Pro Leu Thr Val Lys Cys Thr Tyr Ser Val Ser Gly
370 375 380
Asn Pro Tyr Leu Phe Trp Tyr Val Gln Tyr Pro Asn Arg Gly Leu Gln
385 390 395 400
Phe Leu Leu Lys Tyr Ile Thr Gly Asp Asn Leu Val Lys Gly Ser Tyr
405 410 415
Gly Phe Glu Ala Glu Phe Asn Lys Ser Gln Thr Ser Phe His Leu Lys
420 425 430
Lys Pro Ser Ala Leu Val Ser Asp Ser Ala Leu Tyr Phe Cys Ala Val
435 440 445
Arg Asp Phe Gly Ser Gly Thr Tyr Lys Tyr Ile Phe Gly Thr Gly Thr
450 455 460
Arg Leu Lys Val Leu Ala Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr
465 470 475 480
Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr
485 490 495
Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val
500 505 510
Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys
515 520 525
Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala
530 535 540
Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser
545 550 555 560
Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr
565 570 575
Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile
580 585 590
Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu
595 600 605
Trp Ser Ser
610
<210> 389
<211> 495
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 15 - альфа нативный
<400> 389
gaattcttct ctcaccatgc caggttcacc tcacagtaca gagtcctgaa aataaagaag 60
aagatttttt tttatctaga aaaggaacca aacatgtcac tttctagcct gctgaaggtg 120
gtcacagctt cactgtggct aggacctggc attgcccaga agataactca aacccaacca 180
ggaatgttcg tgcaggaaaa ggaggctgtg actctggact gcacatatga caccagtgat 240
caaagttatg gtctattctg gtacaagcag cccagcagtg gggaaatgat ttttcttatt 300
tatcaggggt cttatgacga gcaaaatgca acagaaggtc gctactcatt gaatttccag 360
aaggcaagaa aatccgccaa ccttgtcatc tccgcttcac aactggggga ctcagcaatg 420
tatttctgtg caatgaagcc tggtggctac aataagctga tttttggagc agggaccagg 480
ctggctgtac accca 495
<210> 390
<211> 455
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 15 - бета нативный
<400> 390
gatgctcaca gagggcctgg tctagaatat tccacatctg ctctcactct gccatggact 60
cctggacctt ctgctgtgtg tccctttgca tcctggtagc gaagcataca gatgctggag 120
ttatccagtc accccgccat gaggtgacag agatgggaca agaagtgact ctgagatgta 180
aaccaatttc aggccacaac tcccttttct ggtacagaca gaccatgatg cggggactgg 240
agttgctcat ttactttaac aacaacgttc cgatagatga ttcagggatg cccgaggatc 300
gattctcagc taagatgcct aatgcatcat tctccactct gaagatccag ccctcagaac 360
ccagggactc agctgtgtac ttctgtgcca gcagtttggt cggcaggagt cggaccgagc 420
agtacttcgg gccgggcacc aggctcacgg tcaca 455
<210> 391
<211> 610
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 15
полноразмерная последовательность, нативный
<400> 391
Met Asp Ser Trp Thr Phe Cys Cys Val Ser Leu Cys Ile Leu Val Ala
1 5 10 15
Lys His Thr Asp Ala Gly Val Ile Gln Ser Pro Arg His Glu Val Thr
20 25 30
Glu Met Gly Gln Glu Val Thr Leu Arg Cys Lys Pro Ile Ser Gly His
35 40 45
Asn Ser Leu Phe Trp Tyr Arg Gln Thr Met Met Arg Gly Leu Glu Leu
50 55 60
Leu Ile Tyr Phe Asn Asn Asn Val Pro Ile Asp Asp Ser Gly Met Pro
65 70 75 80
Glu Asp Arg Phe Ser Ala Lys Met Pro Asn Ala Ser Phe Ser Thr Leu
85 90 95
Lys Ile Gln Pro Ser Glu Pro Arg Asp Ser Ala Val Tyr Phe Cys Ala
100 105 110
Ser Ser Leu Val Gly Arg Ser Arg Thr Glu Gln Tyr Phe Gly Pro Gly
115 120 125
Thr Arg Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu
130 135 140
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
145 150 155 160
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu
165 170 175
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr
180 185 190
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
195 200 205
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
210 215 220
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
225 230 235 240
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
245 250 255
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr
260 265 270
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
275 280 285
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
290 295 300
Met Val Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe
305 310 315 320
Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met
325 330 335
Ser Leu Ser Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu Gly
340 345 350
Pro Gly Ile Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val
355 360 365
Gln Glu Lys Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp
370 375 380
Gln Ser Tyr Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met
385 390 395 400
Ile Phe Leu Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu
405 410 415
Gly Arg Tyr Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu
420 425 430
Val Ile Ser Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala
435 440 445
Met Lys Pro Gly Gly Tyr Asn Lys Leu Ile Phe Gly Ala Gly Thr Arg
450 455 460
Leu Ala Val His Pro Tyr Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln
465 470 475 480
Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp
485 490 495
Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr
500 505 510
Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser
515 520 525
Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn
530 535 540
Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro
545 550 555 560
Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp
565 570 575
Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu
580 585 590
Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp
595 600 605
Ser Ser
610
<210> 392
<211> 604
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 16 полноразмерная последовательность, нативный
<400> 392
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Ser Leu Trp Gly Arg Ser Asn Gln Pro Gln His Phe Gly Asp Gly Thr
115 120 125
Arg Leu Ser Ile Leu Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu
305 310 315 320
Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Glu Thr Leu
325 330 335
Leu Gly Leu Leu Ile Leu Trp Leu Gln Leu Gln Trp Val Ser Ser Lys
340 345 350
Gln Glu Val Thr Gln Ile Pro Ala Ala Leu Ser Val Pro Glu Gly Glu
355 360 365
Asn Leu Val Leu Asn Cys Ser Phe Thr Asp Ser Ala Ile Tyr Asn Leu
370 375 380
Gln Trp Phe Arg Gln Asp Pro Gly Lys Gly Leu Thr Ser Leu Leu Leu
385 390 395 400
Ile Gln Ser Ser Gln Arg Glu Gln Thr Ser Gly Arg Leu Asn Ala Ser
405 410 415
Leu Asp Lys Ser Ser Gly Arg Ser Thr Leu Tyr Ile Ala Ala Ser Gln
420 425 430
Pro Gly Asp Ser Ala Thr Tyr Leu Cys Ala Val Arg Pro Ala Asn Asn
435 440 445
Asn Asp Met Arg Phe Gly Ala Gly Thr Arg Leu Thr Val Lys Pro Asn
450 455 460
Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser
465 470 475 480
Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn
485 490 495
Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr Val
500 505 510
Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp
515 520 525
Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile
530 535 540
Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val
545 550 555 560
Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln
565 570 575
Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly
580 585 590
Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600
<210> 393
<211> 607
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 17 полноразмерная последовательность, нативный
<400> 393
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Ser Leu Trp Gly Arg Ser Asn Gln Pro Gln His Phe Gly Asp Gly Thr
115 120 125
Arg Leu Ser Ile Leu Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu
305 310 315 320
Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Ser Leu Ser
325 330 335
Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu Gly Pro Gly Ile
340 345 350
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
355 360 365
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Pro Ser Tyr
370 375 380
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
385 390 395 400
Ile Tyr Gln Gly Ser Tyr Asp Gln Gln Asn Ala Thr Glu Gly Arg Tyr
405 410 415
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
420 425 430
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Arg Glu
435 440 445
Gly Arg Gly Asp Lys Ile Ile Phe Gly Lys Gly Thr Arg Leu His Ile
450 455 460
Leu Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp
465 470 475 480
Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser
485 490 495
Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp
500 505 510
Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala
515 520 525
Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn
530 535 540
Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser
545 550 555 560
Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu
565 570 575
Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys
580 585 590
Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600 605
<210> 394
<211> 607
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 18 полноразмерная последовательность, нативный
<400> 394
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Ser Phe Trp Gly Arg Ser Asn Ser Pro Leu His Phe Gly Asn Gly Thr
115 120 125
Arg Leu Thr Val Thr Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu
305 310 315 320
Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Glu Thr Leu
325 330 335
Leu Gly Leu Leu Ile Leu Trp Leu Gln Leu Gln Trp Val Ser Ser Lys
340 345 350
Gln Glu Val Thr Gln Ile Pro Ala Ala Leu Ser Val Pro Glu Gly Glu
355 360 365
Asn Leu Val Leu Asn Cys Ser Phe Thr Asp Ser Ala Ile Tyr Asn Leu
370 375 380
Gln Trp Phe Arg Gln Asp Pro Gly Lys Gly Leu Thr Ser Leu Leu Leu
385 390 395 400
Ile Gln Ser Ser Gln Arg Glu Gln Thr Ser Gly Arg Leu Asn Ala Ser
405 410 415
Leu Asp Lys Ser Ser Gly Arg Ser Thr Leu Tyr Ile Ala Ala Ser Gln
420 425 430
Pro Gly Asp Ser Ala Thr Tyr Leu Cys Ala Glu Gly Asn Ala Gly Gly
435 440 445
Thr Ser Tyr Gly Lys Leu Thr Phe Gly Gln Gly Thr Ile Leu Thr Val
450 455 460
His Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp
465 470 475 480
Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser
485 490 495
Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp
500 505 510
Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala
515 520 525
Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn
530 535 540
Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser
545 550 555 560
Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu
565 570 575
Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys
580 585 590
Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600 605
<210> 395
<211> 611
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 19 полноразмерная последовательность, нативный
<400> 395
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Ser Ser Trp Gly Gln Ser Thr Gly Glu Gln Tyr Phe Gly Pro Gly Thr
115 120 125
Arg Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser
305 310 315 320
Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Ser
325 330 335
Leu Ser Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu Gly Pro
340 345 350
Gly Ile Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln
355 360 365
Glu Lys Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln
370 375 380
Ser Tyr Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile
385 390 395 400
Phe Leu Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly
405 410 415
Arg Tyr Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val
420 425 430
Ile Ser Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met
435 440 445
Arg Glu Asn Thr Gly Thr Ala Ser Lys Leu Thr Phe Gly Thr Gly Thr
450 455 460
Arg Leu Gln Val Thr Leu Asp Ile Gln Asn Pro Asp Pro Ala Val Tyr
465 470 475 480
Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr
485 490 495
Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val
500 505 510
Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys
515 520 525
Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala
530 535 540
Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser
545 550 555 560
Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr
565 570 575
Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile
580 585 590
Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu
595 600 605
Trp Ser Ser
610
<210> 396
<211> 608
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 20 полноразмерная последовательность, нативный
<400> 396
Met Leu Leu Leu Leu Leu Leu Leu Gly Pro Gly Ser Gly Leu Gly Ala
1 5 10 15
Val Val Ser Gln His Pro Ser Arg Val Ile Cys Lys Ser Gly Thr Ser
20 25 30
Val Lys Ile Glu Cys Arg Ser Leu Asp Phe Gln Ala Thr Thr Met Phe
35 40 45
Trp Tyr Arg Gln Phe Pro Lys Gln Ser Leu Met Leu Met Ala Thr Ser
50 55 60
Asn Glu Gly Ser Lys Ala Thr Tyr Glu Gln Gly Val Glu Lys Asp Lys
65 70 75 80
Phe Leu Ile Asn His Ala Ser Leu Thr Leu Ser Thr Leu Thr Val Thr
85 90 95
Ser Ala His Pro Glu Asp Ser Ser Phe Tyr Ile Cys Ser Ala Ser Ser
100 105 110
Leu Ala Arg Arg Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu
115 120 125
Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val
130 135 140
Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu
145 150 155 160
Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp
165 170 175
Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln
180 185 190
Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser
195 200 205
Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His
210 215 220
Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp
225 230 235 240
Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala
245 250 255
Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly
260 265 270
Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr
275 280 285
Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys
290 295 300
Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu
305 310 315 320
Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Asn Met Leu
325 330 335
Thr Ala Ser Leu Leu Arg Ala Val Ile Ala Ser Ile Cys Val Val Ser
340 345 350
Ser Met Ala Gln Lys Val Thr Gln Ala Gln Thr Glu Ile Ser Val Val
355 360 365
Glu Lys Glu Asp Val Thr Leu Asp Cys Val Tyr Glu Thr Arg Asp Thr
370 375 380
Thr Tyr Tyr Leu Phe Trp Tyr Lys Gln Pro Pro Ser Gly Glu Leu Val
385 390 395 400
Phe Leu Ile Arg Arg Asn Ser Phe Asp Glu Gln Asn Glu Ile Ser Gly
405 410 415
Arg Tyr Ser Trp Asn Phe Gln Lys Ser Thr Ser Ser Phe Asn Phe Thr
420 425 430
Ile Thr Ala Ser Gln Val Val Asp Ser Ala Val Tyr Phe Cys Ala Leu
435 440 445
Trp Thr Gly Ala Asn Asn Leu Phe Phe Gly Thr Gly Thr Arg Leu Thr
450 455 460
Val Ile Pro Tyr Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg
465 470 475 480
Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp
485 490 495
Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr
500 505 510
Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser
515 520 525
Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe
530 535 540
Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser
545 550 555 560
Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn
565 570 575
Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu
580 585 590
Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600 605
<210> 397
<211> 625
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 21 полноразмерная последовательность, нативный
<400> 397
Met His Arg Pro Arg Arg Pro Leu His Pro Val Ala Pro Ala Met Ser
1 5 10 15
Ile Gly Leu Leu Cys Cys Val Ala Phe Ser Leu Leu Trp Ala Ser Pro
20 25 30
Val Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Gln Val Leu Lys Thr
35 40 45
Gly Gln Ser Met Thr Leu Gln Cys Ala Gln Asp Met Asn His Asn Ser
50 55 60
Met Tyr Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu Ile Tyr
65 70 75 80
Tyr Ser Ala Ser Glu Gly Thr Thr Asp Lys Gly Glu Val Pro Asn Gly
85 90 95
Tyr Asn Val Ser Arg Leu Asn Lys Arg Glu Phe Ser Leu Arg Leu Glu
100 105 110
Ser Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser Arg Pro
115 120 125
Trp Gly Asn Gln Asn Thr Glu Ala Phe Phe Gly Gln Gly Thr Arg Leu
130 135 140
Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val
145 150 155 160
Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu
165 170 175
Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp
180 185 190
Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln
195 200 205
Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser
210 215 220
Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His
225 230 235 240
Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp
245 250 255
Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala
260 265 270
Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly
275 280 285
Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr
290 295 300
Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys
305 310 315 320
Arg Lys Asp Phe Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu Lys Gln
325 330 335
Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Ser Leu Ser Ser Leu
340 345 350
Leu Lys Val Val Thr Ala Ser Leu Trp Leu Gly Pro Gly Ile Ala Gln
355 360 365
Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys Glu Ala
370 375 380
Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln Ser Tyr Gly Leu
385 390 395 400
Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu Ile Tyr
405 410 415
Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly Arg Tyr Ser Leu
420 425 430
Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser Ala Ser
435 440 445
Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Arg Glu Gly Arg
450 455 460
Val Thr Gly Gly Gly Asn Lys Leu Thr Phe Gly Thr Gly Thr Gln Leu
465 470 475 480
Lys Val Glu Leu Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu
485 490 495
Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe
500 505 510
Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile
515 520 525
Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn
530 535 540
Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala
545 550 555 560
Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu
565 570 575
Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr
580 585 590
Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu
595 600 605
Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser
610 615 620
Ser
625
<210> 398
<211> 617
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 22 полноразмерная последовательность, нативный
<400> 398
Met Gly Pro Gly Leu Leu Cys Trp Val Leu Leu Cys Leu Leu Gly Ala
1 5 10 15
Gly Pro Val Asp Ala Gly Val Thr Gln Ser Pro Thr His Leu Ile Lys
20 25 30
Thr Arg Gly Gln His Val Thr Leu Arg Cys Ser Pro Ile Ser Gly His
35 40 45
Lys Ser Val Ser Trp Tyr Gln Gln Val Leu Gly Gln Gly Pro Gln Phe
50 55 60
Ile Phe Gln Tyr Tyr Glu Lys Glu Glu Arg Gly Arg Gly Asn Phe Pro
65 70 75 80
Asp Arg Phe Ser Ala Arg Gln Phe Pro Asn Tyr Ser Ser Glu Leu Asn
85 90 95
Val Asn Ala Leu Leu Leu Gly Asp Ser Ala Leu Tyr Leu Cys Ala Ser
100 105 110
Ser Arg Thr Glu Asn Tyr Gly Tyr Thr Phe Gly Ser Gly Thr Arg Leu
115 120 125
Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val
130 135 140
Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu
145 150 155 160
Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp
165 170 175
Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln
180 185 190
Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser
195 200 205
Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His
210 215 220
Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp
225 230 235 240
Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala
245 250 255
Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly
260 265 270
Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr
275 280 285
Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys
290 295 300
Arg Lys Asp Phe Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu Lys Gln
305 310 315 320
Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Ala Gln Glu Leu Gly
325 330 335
Met Gln Cys Gln Ala Arg Gly Ile Leu Gln Gln Met Trp Gly Val Phe
340 345 350
Leu Leu Tyr Val Ser Met Lys Met Gly Gly Thr Thr Gly Gln Asn Ile
355 360 365
Asp Gln Pro Thr Glu Met Thr Ala Thr Glu Gly Ala Ile Val Gln Ile
370 375 380
Asn Cys Thr Tyr Gln Thr Ser Gly Phe Asn Gly Leu Phe Trp Tyr Gln
385 390 395 400
Gln His Ala Gly Glu Ala Pro Thr Phe Leu Ser Tyr Asn Val Leu Asp
405 410 415
Gly Leu Glu Glu Lys Gly Arg Phe Ser Ser Phe Leu Ser Arg Ser Lys
420 425 430
Gly Tyr Ser Tyr Leu Leu Leu Lys Glu Leu Gln Met Lys Asp Ser Ala
435 440 445
Ser Tyr Leu Cys Ala Val Arg Ala Arg Met Asp Ser Asn Tyr Gln Leu
450 455 460
Ile Trp Gly Ala Gly Thr Lys Leu Ile Ile Lys Pro Asp Ile Gln Asn
465 470 475 480
Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys
485 490 495
Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln
500 505 510
Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr Val Leu Asp Met
515 520 525
Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys
530 535 540
Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu
545 550 555 560
Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val
565 570 575
Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser
580 585 590
Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu
595 600 605
Leu Met Thr Leu Arg Leu Trp Ser Ser
610 615
<210> 399
<211> 609
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 23 полноразмерная последовательность, нативный
<400> 399
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Ser Pro Trp Gly Gln Ser Asn Gln Pro Gln His Phe Gly Asp Gly Thr
115 120 125
Arg Leu Ser Ile Leu Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu
305 310 315 320
Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Ser Leu Ser
325 330 335
Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu Gly Pro Gly Ile
340 345 350
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
355 360 365
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln Ser Tyr
370 375 380
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
385 390 395 400
Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly Arg Tyr
405 410 415
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
420 425 430
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Ser Pro
435 440 445
Pro Gly Gly Ser Ala Arg Gln Leu Thr Phe Gly Ser Gly Thr Gln Leu
450 455 460
Thr Val Leu Pro Asp Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu
465 470 475 480
Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe
485 490 495
Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile
500 505 510
Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn
515 520 525
Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala
530 535 540
Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu
545 550 555 560
Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr
565 570 575
Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu
580 585 590
Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser
595 600 605
Ser
<210> 400
<211> 612
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 24 полноразмерная последовательность, нативный
<400> 400
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Ser Pro Phe Gly Arg Gly Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr
115 120 125
Arg Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser
305 310 315 320
Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Ser
325 330 335
Leu Ser Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu Gly Pro
340 345 350
Gly Ile Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln
355 360 365
Glu Lys Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln
370 375 380
Ser Tyr Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile
385 390 395 400
Phe Leu Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly
405 410 415
Arg Tyr Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val
420 425 430
Ile Ser Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met
435 440 445
Arg Glu Gly Arg Gly Asp Ser Trp Gly Lys Leu Gln Phe Gly Ala Gly
450 455 460
Thr Gln Val Val Val Thr Pro Asp Ile Gln Asn Pro Asp Pro Ala Val
465 470 475 480
Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe
485 490 495
Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp
500 505 510
Val Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe
515 520 525
Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys
530 535 540
Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro
545 550 555 560
Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu
565 570 575
Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg
580 585 590
Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg
595 600 605
Leu Trp Ser Ser
610
<210> 401
<211> 611
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 25 полноразмерная последовательность, нативный
<400> 401
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Ser Leu Trp Gly Gln Ser Asn Gln Pro Gln His Phe Gly Asp Gly Thr
115 120 125
Arg Leu Ser Ile Leu Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu
305 310 315 320
Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Ser Leu Ser
325 330 335
Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu Gly Pro Gly Ile
340 345 350
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
355 360 365
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln Ser Tyr
370 375 380
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
385 390 395 400
Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly Arg Tyr
405 410 415
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
420 425 430
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Arg Glu
435 440 445
Gly Ser Leu Thr Gly Gly Gly Asn Lys Leu Thr Phe Gly Thr Gly Thr
450 455 460
Gln Leu Lys Val Glu Leu Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr
465 470 475 480
Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr
485 490 495
Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val
500 505 510
Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys
515 520 525
Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala
530 535 540
Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser
545 550 555 560
Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr
565 570 575
Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile
580 585 590
Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu
595 600 605
Trp Ser Ser
610
<210> 402
<211> 610
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 26 полноразмерная последовательность, нативный
<400> 402
Met Ser Leu Gly Leu Leu Cys Cys Gly Ala Phe Ser Leu Leu Trp Ala
1 5 10 15
Gly Pro Val Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Arg Val Leu
20 25 30
Lys Thr Gly Gln Ser Met Thr Leu Leu Cys Ala Gln Asp Met Asn His
35 40 45
Glu Tyr Met Tyr Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu
50 55 60
Ile His Tyr Ser Val Gly Glu Gly Thr Thr Ala Lys Gly Glu Val Pro
65 70 75 80
Asp Gly Tyr Asn Val Ser Arg Leu Lys Lys Gln Asn Phe Leu Leu Gly
85 90 95
Leu Glu Ser Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser
100 105 110
Ser Tyr Tyr Ala Ser Gly Arg Asn Tyr Glu Gln Tyr Phe Gly Pro Gly
115 120 125
Thr Arg Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu
130 135 140
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
145 150 155 160
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu
165 170 175
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr
180 185 190
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
195 200 205
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
210 215 220
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
225 230 235 240
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
245 250 255
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr
260 265 270
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
275 280 285
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
290 295 300
Met Val Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe
305 310 315 320
Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met
325 330 335
Ser Leu Ser Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu Gly
340 345 350
Pro Gly Ile Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val
355 360 365
Gln Glu Lys Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp
370 375 380
Gln Ser Tyr Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met
385 390 395 400
Ile Phe Leu Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu
405 410 415
Gly Arg Tyr Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu
420 425 430
Val Ile Ser Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala
435 440 445
Met Arg Asp Ala Arg Asn Asn Asp Met Arg Phe Gly Ala Gly Thr Arg
450 455 460
Leu Thr Val Lys Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln
465 470 475 480
Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp
485 490 495
Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr
500 505 510
Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser
515 520 525
Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn
530 535 540
Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro
545 550 555 560
Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp
565 570 575
Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu
580 585 590
Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp
595 600 605
Ser Ser
610
<210> 403
<211> 618
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 27 полноразмерная последовательность, нативный
<400> 403
Met His Arg Pro Arg Arg Pro Leu His Pro Val Ala Pro Ala Met Ser
1 5 10 15
Ile Gly Leu Leu Cys Cys Val Ala Phe Ser Leu Leu Trp Ala Ser Pro
20 25 30
Val Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Gln Val Leu Lys Thr
35 40 45
Gly Gln Ser Met Thr Leu Gln Cys Ala Gln Asp Met Asn His Asn Ser
50 55 60
Met Tyr Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu Ile Tyr
65 70 75 80
Tyr Ser Ala Ser Glu Gly Thr Thr Asp Lys Gly Glu Val Pro Asn Gly
85 90 95
Tyr Asn Val Ser Arg Leu Asn Lys Arg Glu Phe Ser Leu Arg Leu Glu
100 105 110
Ser Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser Ser Glu
115 120 125
Phe Gly Ser Leu Asn Glu Lys Leu Phe Phe Gly Ser Gly Thr Gln Leu
130 135 140
Ser Val Leu Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val
145 150 155 160
Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu
165 170 175
Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp
180 185 190
Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln
195 200 205
Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser
210 215 220
Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His
225 230 235 240
Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp
245 250 255
Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala
260 265 270
Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly
275 280 285
Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr
290 295 300
Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys
305 310 315 320
Arg Lys Asp Phe Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu Lys Gln
325 330 335
Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Lys Leu Val Thr Ser
340 345 350
Ile Thr Val Leu Leu Ser Leu Gly Ile Met Gly Asp Ala Lys Thr Thr
355 360 365
Gln Pro Asn Ser Met Glu Ser Asn Glu Glu Glu Pro Val His Leu Pro
370 375 380
Cys Asn His Ser Thr Ile Ser Gly Thr Asp Tyr Ile His Trp Tyr Arg
385 390 395 400
Gln Leu Pro Ser Gln Gly Pro Glu Tyr Val Ile His Gly Leu Thr Ser
405 410 415
Asn Val Asn Asn Arg Met Ala Ser Leu Ala Ile Ala Glu Asp Arg Lys
420 425 430
Ser Ser Thr Leu Ile Leu His Arg Ala Thr Leu Arg Asp Ala Ala Val
435 440 445
Tyr Tyr Cys Ile Leu Arg Val Pro Pro Gln Ser Gly Gly Tyr Gln Lys
450 455 460
Val Thr Phe Gly Thr Gly Thr Lys Leu Gln Val Ile Pro Asn Ile Gln
465 470 475 480
Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp
485 490 495
Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser
500 505 510
Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr Val Leu Asp
515 520 525
Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn
530 535 540
Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro
545 550 555 560
Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu
565 570 575
Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu
580 585 590
Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn
595 600 605
Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
610 615
<210> 404
<211> 608
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 28 полноразмерная последовательность, нативный
<400> 404
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Ser Leu Trp Gly Arg Ser Ser Gly Asn Thr Ile Tyr Phe Gly Glu Gly
115 120 125
Ser Trp Leu Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu
130 135 140
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
145 150 155 160
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu
165 170 175
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr
180 185 190
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
195 200 205
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
210 215 220
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
225 230 235 240
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
245 250 255
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr
260 265 270
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
275 280 285
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
290 295 300
Met Val Lys Arg Lys Asp Phe Gly Ser Gly Ala Thr Asn Phe Ser Leu
305 310 315 320
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Met Ile
325 330 335
Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu Ser Trp Val
340 345 350
Trp Ser Gln Arg Lys Glu Val Glu Gln Asp Pro Gly Pro Phe Asn Val
355 360 365
Pro Glu Gly Ala Thr Val Ala Phe Asn Cys Thr Tyr Ser Asn Ser Ala
370 375 380
Ser Gln Ser Phe Phe Trp Tyr Arg Gln Asp Cys Arg Lys Glu Pro Lys
385 390 395 400
Leu Leu Met Ser Val Tyr Ser Ser Gly Asn Glu Asp Gly Arg Phe Thr
405 410 415
Ala Gln Leu Asn Arg Ala Ser Gln Tyr Ile Ser Leu Leu Ile Arg Asp
420 425 430
Ser Lys Leu Ser Asp Ser Ala Thr Tyr Leu Cys Val Val Arg Gly Gly
435 440 445
Gly Thr Ser Tyr Gly Lys Leu Thr Phe Gly Gln Gly Thr Ile Leu Thr
450 455 460
Val His Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg
465 470 475 480
Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp
485 490 495
Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr
500 505 510
Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser
515 520 525
Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe
530 535 540
Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser
545 550 555 560
Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn
565 570 575
Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu
580 585 590
Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600 605
<210> 405
<211> 611
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 29 полноразмерная последовательность, нативный
<400> 405
Met Ser Asn Gln Val Leu Cys Cys Val Val Leu Cys Leu Leu Gly Ala
1 5 10 15
Asn Thr Val Asp Gly Gly Ile Thr Gln Ser Pro Lys Tyr Leu Phe Arg
20 25 30
Lys Glu Gly Gln Asn Val Thr Leu Ser Cys Glu Gln Asn Leu Asn His
35 40 45
Asp Ala Met Tyr Trp Tyr Arg Gln Asp Pro Gly Gln Gly Leu Arg Leu
50 55 60
Ile Tyr Tyr Ser Gln Ile Val Asn Asp Phe Gln Lys Gly Asp Ile Ala
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Ser Phe Pro Leu Thr
85 90 95
Val Thr Ser Ala Gln Lys Asn Pro Thr Ala Phe Tyr Leu Cys Ala Ser
100 105 110
Ser Pro Trp Gly Arg Ala Thr Asn Glu Gln Phe Phe Gly Pro Gly Thr
115 120 125
Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser
305 310 315 320
Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Ser
325 330 335
Leu Ser Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu Gly Pro
340 345 350
Gly Ile Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln
355 360 365
Glu Lys Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln
370 375 380
Ser Tyr Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile
385 390 395 400
Phe Leu Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly
405 410 415
Arg Tyr Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val
420 425 430
Ile Ser Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met
435 440 445
Arg Leu Asn Thr Gly Thr Ala Ser Lys Leu Thr Phe Gly Thr Gly Thr
450 455 460
Arg Leu Gln Val Thr Leu Asp Ile Gln Asn Pro Asp Pro Ala Val Tyr
465 470 475 480
Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr
485 490 495
Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val
500 505 510
Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys
515 520 525
Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala
530 535 540
Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser
545 550 555 560
Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr
565 570 575
Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile
580 585 590
Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu
595 600 605
Trp Ser Ser
610
<210> 406
<211> 605
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 30 полноразмерная последовательность, нативный
<400> 406
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ser Ser Arg Gln Pro Ser Ser Gly Asn Thr Ile Tyr Phe Gly Glu Gly
115 120 125
Ser Trp Leu Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu
130 135 140
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
145 150 155 160
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu
165 170 175
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr
180 185 190
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
195 200 205
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
210 215 220
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
225 230 235 240
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
245 250 255
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr
260 265 270
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
275 280 285
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
290 295 300
Met Val Lys Arg Lys Asp Phe Gly Ser Gly Ala Thr Asn Phe Ser Leu
305 310 315 320
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Arg Leu
325 330 335
Val Ala Arg Val Thr Val Phe Leu Thr Phe Gly Thr Ile Ile Asp Ala
340 345 350
Lys Thr Thr Gln Pro Pro Ser Met Asp Cys Ala Glu Gly Arg Ala Ala
355 360 365
Asn Leu Pro Cys Asn His Ser Thr Ile Ser Gly Asn Glu Tyr Val Tyr
370 375 380
Trp Tyr Arg Gln Ile His Ser Gln Gly Pro Gln Tyr Ile Ile His Gly
385 390 395 400
Leu Lys Asn Asn Glu Thr Asn Glu Met Ala Ser Leu Ile Ile Thr Glu
405 410 415
Asp Arg Lys Ser Ser Thr Leu Ile Leu Pro His Ala Thr Leu Arg Asp
420 425 430
Thr Ala Val Tyr Tyr Cys Ile Val Arg Gly Thr Ser Val Leu Gln Gly
435 440 445
Asn Glu Lys Leu Thr Phe Gly Thr Gly Thr Arg Leu Thr Ile Ile Pro
450 455 460
Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys
465 470 475 480
Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr
485 490 495
Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr
500 505 510
Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala
515 520 525
Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser
530 535 540
Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp
545 550 555 560
Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe
565 570 575
Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala
580 585 590
Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600 605
<210> 407
<211> 614
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 31 полноразмерная последовательность, нативный
<400> 407
Met Gly Thr Arg Leu Leu Phe Trp Val Ala Phe Cys Leu Leu Gly Ala
1 5 10 15
Asp His Thr Gly Ala Gly Val Ser Gln Ser Pro Ser Asn Lys Val Thr
20 25 30
Glu Lys Gly Lys Asp Val Glu Leu Arg Cys Asp Pro Ile Ser Gly His
35 40 45
Thr Ala Leu Tyr Trp Tyr Arg Gln Ser Leu Gly Gln Gly Leu Glu Phe
50 55 60
Leu Ile Tyr Phe Gln Gly Asn Ser Ala Pro Asp Lys Ser Gly Leu Pro
65 70 75 80
Ser Asp Arg Phe Ser Ala Glu Arg Thr Gly Gly Ser Val Ser Thr Leu
85 90 95
Thr Ile Gln Arg Thr Gln Gln Glu Asp Ser Ala Val Tyr Leu Cys Ala
100 105 110
Ser Ser Arg Phe Leu Gly Ser Thr Asp Thr Gln Tyr Phe Gly Pro Gly
115 120 125
Thr Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu
130 135 140
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
145 150 155 160
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu
165 170 175
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr
180 185 190
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
195 200 205
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
210 215 220
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
225 230 235 240
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
245 250 255
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr
260 265 270
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
275 280 285
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
290 295 300
Met Val Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe
305 310 315 320
Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met
325 330 335
Ala Met Leu Leu Gly Ala Ser Val Leu Ile Leu Trp Leu Gln Pro Asp
340 345 350
Trp Val Asn Ser Gln Gln Lys Asn Asp Asp Gln Gln Val Lys Gln Asn
355 360 365
Ser Pro Ser Leu Ser Val Gln Glu Gly Arg Ile Ser Ile Leu Asn Cys
370 375 380
Asp Tyr Thr Asn Ser Met Phe Asp Tyr Phe Leu Trp Tyr Lys Lys Tyr
385 390 395 400
Pro Ala Glu Gly Pro Thr Phe Leu Ile Ser Ile Ser Ser Ile Lys Asp
405 410 415
Lys Asn Glu Asp Gly Arg Phe Thr Val Phe Leu Asn Lys Ser Ala Lys
420 425 430
His Leu Ser Leu His Ile Val Pro Ser Gln Pro Gly Asp Ser Ala Val
435 440 445
Tyr Phe Cys Ala Ala Ser Glu Arg Gly Thr Tyr Lys Tyr Ile Phe Gly
450 455 460
Thr Gly Thr Arg Leu Lys Val Leu Ala Asn Ile Gln Asn Pro Asp Pro
465 470 475 480
Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys
485 490 495
Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp
500 505 510
Ser Asp Val Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met
515 520 525
Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe
530 535 540
Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe
545 550 555 560
Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser
565 570 575
Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly
580 585 590
Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr
595 600 605
Leu Arg Leu Trp Ser Ser
610
<210> 408
<211> 602
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 32 полноразмерная последовательность, нативный
<400> 408
Met Gly Phe Arg Leu Leu Cys Cys Val Ala Phe Cys Leu Leu Gly Ala
1 5 10 15
Gly Pro Val Asp Ser Gly Val Thr Gln Thr Pro Lys His Leu Ile Thr
20 25 30
Ala Thr Gly Gln Arg Val Thr Leu Arg Cys Ser Pro Arg Ser Gly Asp
35 40 45
Leu Ser Val Tyr Trp Tyr Gln Gln Ser Leu Asp Gln Gly Leu Gln Phe
50 55 60
Leu Ile Gln Tyr Tyr Asn Gly Glu Glu Arg Ala Lys Gly Asn Ile Leu
65 70 75 80
Glu Arg Phe Ser Ala Gln Gln Phe Pro Asp Leu His Ser Glu Leu Asn
85 90 95
Leu Ser Ser Leu Glu Leu Gly Asp Ser Ala Leu Tyr Phe Cys Ala Ser
100 105 110
Ser Val Gly Gly Asp His Ser Asp Glu Gln Phe Phe Gly Pro Gly Thr
115 120 125
Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser
305 310 315 320
Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Val
325 330 335
Leu Lys Phe Ser Val Ser Ile Leu Trp Ile Gln Leu Ala Trp Val Ser
340 345 350
Thr Gln Leu Leu Glu Gln Ser Pro Gln Phe Leu Ser Ile Gln Glu Gly
355 360 365
Glu Asn Leu Thr Val Tyr Cys Asn Ser Ser Ser Val Phe Ser Ser Leu
370 375 380
Gln Trp Tyr Arg Gln Glu Pro Gly Glu Gly Pro Val Leu Leu Val Thr
385 390 395 400
Val Val Thr Gly Gly Glu Val Lys Lys Leu Lys Arg Leu Thr Phe Gln
405 410 415
Phe Gly Asp Ala Arg Lys Asp Ser Ser Leu His Ile Thr Ala Ala Gln
420 425 430
Pro Gly Asp Thr Gly Leu Tyr Leu Cys Ala Gly Gly Ser Asn Tyr Lys
435 440 445
Leu Thr Phe Gly Lys Gly Thr Leu Leu Thr Val Asn Pro Asn Ile Gln
450 455 460
Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp
465 470 475 480
Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser
485 490 495
Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr Val Leu Asp
500 505 510
Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn
515 520 525
Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro
530 535 540
Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu
545 550 555 560
Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu
565 570 575
Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn
580 585 590
Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600
<210> 409
<211> 609
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 33 полноразмерная последовательность, нативный
<400> 409
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ser Thr Pro Arg Asp Thr Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser Leu
305 310 315 320
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Met Lys
325 330 335
Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu Ser Trp Val
340 345 350
Trp Ser Gln Gln Lys Glu Val Glu Gln Asn Ser Gly Pro Leu Ser Val
355 360 365
Pro Glu Gly Ala Ile Ala Ser Leu Asn Cys Thr Tyr Ser Asp Arg Gly
370 375 380
Ser Gln Ser Phe Phe Trp Tyr Arg Gln Tyr Ser Gly Lys Ser Pro Glu
385 390 395 400
Leu Ile Met Phe Ile Tyr Ser Asn Gly Asp Lys Glu Asp Gly Arg Phe
405 410 415
Thr Ala Gln Leu Asn Lys Ala Ser Gln Tyr Val Ser Leu Leu Ile Arg
420 425 430
Asp Ser Gln Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala Val Asn Ala
435 440 445
His His Thr Gly Gly Phe Lys Thr Ile Phe Gly Ala Gly Thr Arg Leu
450 455 460
Phe Val Lys Ala Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu
465 470 475 480
Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe
485 490 495
Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile
500 505 510
Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn
515 520 525
Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala
530 535 540
Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu
545 550 555 560
Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr
565 570 575
Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu
580 585 590
Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser
595 600 605
Ser
<210> 410
<211> 601
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 34 полноразмерная последовательность, нативный
<400> 410
Met Gly Pro Gly Leu Leu Cys Trp Ala Leu Leu Cys Leu Leu Gly Ala
1 5 10 15
Gly Ser Val Glu Thr Gly Val Thr Gln Ser Pro Thr His Leu Ile Lys
20 25 30
Thr Arg Gly Gln Gln Val Thr Leu Arg Cys Ser Ser Gln Ser Gly His
35 40 45
Asn Thr Val Ser Trp Tyr Gln Gln Ala Leu Gly Gln Gly Pro Gln Phe
50 55 60
Ile Phe Gln Tyr Tyr Arg Glu Glu Glu Asn Gly Arg Gly Asn Phe Pro
65 70 75 80
Pro Arg Phe Ser Gly Leu Gln Phe Pro Asn Tyr Ser Ser Glu Leu Asn
85 90 95
Val Asn Ala Leu Glu Leu Asp Asp Ser Ala Leu Tyr Leu Cys Ala Ser
100 105 110
Ser Ser Tyr Ala Gly Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser Leu
305 310 315 320
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Lys Lys
325 330 335
Leu Leu Ala Met Ile Leu Trp Leu Gln Leu Asp Arg Leu Ser Gly Glu
340 345 350
Leu Lys Val Glu Gln Asn Pro Leu Phe Leu Ser Met Gln Glu Gly Lys
355 360 365
Asn Tyr Thr Ile Tyr Cys Asn Tyr Ser Thr Thr Ser Asp Arg Leu Tyr
370 375 380
Trp Tyr Arg Gln Asp Pro Gly Lys Ser Leu Glu Ser Leu Phe Val Leu
385 390 395 400
Leu Ser Asn Gly Ala Val Lys Gln Glu Gly Arg Leu Met Ala Ser Leu
405 410 415
Asp Thr Lys Ala Arg Leu Ser Thr Leu His Ile Thr Ala Ala Val His
420 425 430
Asp Leu Ser Ala Thr Tyr Phe Cys Ala Val Ser Gly Thr Tyr Lys Tyr
435 440 445
Ile Phe Gly Thr Gly Thr Arg Leu Lys Val Leu Ala Asn Ile Gln Asn
450 455 460
Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys
465 470 475 480
Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln
485 490 495
Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr Val Leu Asp Met
500 505 510
Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys
515 520 525
Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu
530 535 540
Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val
545 550 555 560
Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser
565 570 575
Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu
580 585 590
Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600
<210> 411
<211> 605
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 35 полноразмерная последовательность, нативный
<400> 411
Met Gly Phe Arg Leu Leu Cys Cys Val Ala Phe Cys Leu Leu Gly Ala
1 5 10 15
Gly Pro Val Asp Ser Gly Val Thr Gln Thr Pro Lys His Leu Ile Thr
20 25 30
Ala Thr Gly Gln Arg Val Thr Leu Arg Cys Ser Pro Arg Ser Gly Asp
35 40 45
Leu Ser Val Tyr Trp Tyr Gln Gln Ser Leu Asp Gln Gly Leu Gln Phe
50 55 60
Leu Ile Gln Tyr Tyr Asn Gly Glu Glu Arg Ala Lys Gly Asn Ile Leu
65 70 75 80
Glu Arg Phe Ser Ala Gln Gln Phe Pro Asp Leu His Ser Glu Leu Asn
85 90 95
Leu Ser Ser Leu Glu Leu Gly Asp Ser Ala Leu Tyr Phe Cys Ala Ser
100 105 110
Thr Thr Ser Gly Asp Ser Ser Tyr Asn Glu Gln Phe Phe Gly Pro Gly
115 120 125
Thr Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu
130 135 140
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
145 150 155 160
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu
165 170 175
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr
180 185 190
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
195 200 205
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
210 215 220
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
225 230 235 240
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
245 250 255
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr
260 265 270
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
275 280 285
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
290 295 300
Met Val Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe
305 310 315 320
Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met
325 330 335
Ala Leu Gln Ser Thr Leu Gly Ala Val Trp Leu Gly Leu Leu Leu Asn
340 345 350
Ser Leu Trp Lys Val Ala Glu Ser Lys Asp Gln Val Phe Gln Pro Ser
355 360 365
Thr Val Ala Ser Ser Glu Gly Ala Val Val Glu Ile Phe Cys Asn His
370 375 380
Ser Val Ser Asn Ala Tyr Asn Phe Phe Trp Tyr Leu His Phe Pro Gly
385 390 395 400
Cys Ala Pro Arg Leu Leu Val Lys Gly Ser Lys Pro Ser Gln Gln Gly
405 410 415
Arg Tyr Asn Met Thr Tyr Glu Arg Phe Ser Ser Ser Leu Leu Ile Leu
420 425 430
Gln Val Arg Glu Ala Asp Ala Ala Val Tyr Tyr Cys Ala Val Ala Gly
435 440 445
Asp Tyr Lys Leu Ser Phe Gly Ala Gly Thr Thr Val Thr Val Arg Ala
450 455 460
Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys
465 470 475 480
Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr
485 490 495
Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr
500 505 510
Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala
515 520 525
Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser
530 535 540
Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp
545 550 555 560
Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe
565 570 575
Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala
580 585 590
Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600 605
<210> 412
<211> 607
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 36 полноразмерная последовательность, нативный
<400> 412
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Met Thr Gly Arg Ser Asn Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser Leu
305 310 315 320
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Met Ile
325 330 335
Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu Ser Trp Val
340 345 350
Trp Ser Gln Arg Lys Glu Val Glu Gln Asp Pro Gly Pro Phe Asn Val
355 360 365
Pro Glu Gly Ala Thr Val Ala Phe Asn Cys Thr Tyr Ser Asn Ser Ala
370 375 380
Ser Gln Ser Phe Phe Trp Tyr Arg Gln Asp Cys Arg Lys Glu Pro Lys
385 390 395 400
Leu Leu Met Ser Val Tyr Ser Ser Gly Asn Glu Asp Gly Arg Phe Thr
405 410 415
Ala Gln Leu Asn Arg Ala Ser Gln Tyr Ile Ser Leu Leu Ile Arg Asp
420 425 430
Ser Lys Leu Ser Asp Ser Ala Thr Tyr Leu Cys Val Val Asn Arg Asp
435 440 445
Asn Tyr Gly Gln Asn Phe Val Phe Gly Pro Gly Thr Arg Leu Ser Val
450 455 460
Leu Pro Tyr Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp
465 470 475 480
Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser
485 490 495
Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp
500 505 510
Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala
515 520 525
Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn
530 535 540
Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser
545 550 555 560
Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu
565 570 575
Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys
580 585 590
Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600 605
<210> 413
<211> 603
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 37 полноразмерная последовательность, нативный
<400> 413
Met Gly Thr Arg Leu Leu Cys Trp Val Val Leu Gly Phe Leu Gly Thr
1 5 10 15
Asp His Thr Gly Ala Gly Val Ser Gln Ser Pro Arg Tyr Lys Val Ala
20 25 30
Lys Arg Gly Gln Asp Val Ala Leu Arg Cys Asp Pro Ile Ser Gly His
35 40 45
Val Ser Leu Phe Trp Tyr Gln Gln Ala Leu Gly Gln Gly Pro Glu Phe
50 55 60
Leu Thr Tyr Phe Gln Asn Glu Ala Gln Leu Asp Lys Ser Gly Leu Pro
65 70 75 80
Ser Asp Arg Phe Phe Ala Glu Arg Pro Glu Gly Ser Val Ser Thr Leu
85 90 95
Lys Ile Gln Arg Thr Gln Gln Glu Asp Ser Ala Val Tyr Leu Cys Ala
100 105 110
Ser Ser Leu Leu Leu Gly Ala Tyr Gly Tyr Thr Phe Gly Ser Gly Thr
115 120 125
Arg Leu Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu
305 310 315 320
Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Lys Lys Leu
325 330 335
Leu Ala Met Ile Leu Trp Leu Gln Leu Asp Arg Leu Ser Gly Glu Leu
340 345 350
Lys Val Glu Gln Asn Pro Leu Phe Leu Ser Met Gln Glu Gly Lys Asn
355 360 365
Tyr Thr Ile Tyr Cys Asn Tyr Ser Thr Thr Ser Asp Arg Leu Tyr Trp
370 375 380
Tyr Arg Gln Asp Pro Gly Lys Ser Leu Glu Ser Leu Phe Val Leu Leu
385 390 395 400
Ser Asn Gly Ala Val Lys Gln Glu Gly Arg Leu Met Ala Ser Leu Asp
405 410 415
Thr Lys Ala Arg Leu Ser Thr Leu His Ile Thr Ala Ala Val His Asp
420 425 430
Leu Ser Ala Thr Tyr Phe Cys Ala Gly Tyr Ser Gly Ala Gly Ser Tyr
435 440 445
Gln Leu Thr Phe Gly Lys Gly Thr Lys Leu Ser Val Ile Pro Asn Ile
450 455 460
Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser
465 470 475 480
Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val
485 490 495
Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr Val Leu
500 505 510
Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser
515 520 525
Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile
530 535 540
Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys
545 550 555 560
Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn
565 570 575
Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe
580 585 590
Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600
<210> 414
<211> 612
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 38 полноразмерная последовательность, нативный
<400> 414
Met Gly Pro Gly Leu Leu Cys Trp Val Leu Leu Cys Leu Leu Gly Ala
1 5 10 15
Gly Ser Val Glu Thr Gly Val Thr Gln Ser Pro Thr His Leu Ile Lys
20 25 30
Thr Arg Gly Gln Gln Val Thr Leu Arg Cys Ser Ser Gln Ser Gly His
35 40 45
Asn Thr Val Ser Trp Tyr Gln Gln Ala Leu Gly Gln Gly Pro Gln Phe
50 55 60
Ile Phe Gln Tyr Tyr Arg Glu Glu Glu Asn Gly Arg Gly Asn Phe Pro
65 70 75 80
Pro Arg Phe Ser Gly Leu Gln Phe Pro Asn Tyr Ser Ser Glu Leu Asn
85 90 95
Val Asn Ala Leu Glu Leu Asp Asp Ser Ala Leu Tyr Leu Cys Ala Ser
100 105 110
Ser Leu Val Ala Gly Gly Glu Thr Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Leu Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser Leu
305 310 315 320
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Val Lys
325 330 335
Met Pro Gly Ala Arg Arg Gln Ser Ile Met Lys Arg Ile Leu Gly Ala
340 345 350
Leu Leu Gly Leu Leu Ser Ala Gln Val Cys Cys Val Arg Gly Ile Gln
355 360 365
Val Glu Gln Ser Pro Pro Asp Leu Ile Leu Gln Glu Gly Ala Asn Ser
370 375 380
Thr Leu Arg Cys Asn Phe Ser Asp Ser Val Asn Asn Leu Gln Trp Phe
385 390 395 400
His Gln Asn Pro Trp Gly Gln Leu Ile Asn Leu Phe Tyr Ile Pro Ser
405 410 415
Gly Thr Lys Gln Asn Gly Arg Leu Ser Ala Thr Thr Val Ala Thr Glu
420 425 430
Arg Tyr Ser Leu Leu Tyr Ile Ser Ser Ser Gln Thr Thr Asp Ser Gly
435 440 445
Val Tyr Phe Cys Ala Val Gly Phe Asn Asp Met Arg Phe Gly Ala Gly
450 455 460
Thr Arg Leu Thr Val Lys Pro Asn Ile Gln Asn Pro Asp Pro Ala Val
465 470 475 480
Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe
485 490 495
Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp
500 505 510
Val Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe
515 520 525
Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys
530 535 540
Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro
545 550 555 560
Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu
565 570 575
Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg
580 585 590
Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg
595 600 605
Leu Trp Ser Ser
610
<210> 415
<211> 592
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 39 полноразмерная последовательность, нативный
<400> 415
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ser Thr Pro Arg Asp Arg Gly Lys Glu Ala Phe Phe Gly Gln Gly Thr
115 120 125
Arg Leu Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu
305 310 315 320
Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Gln Leu Thr
325 330 335
Trp Val Ser Gly Gln Gln Leu Asn Gln Ser Pro Gln Ser Met Phe Ile
340 345 350
Gln Glu Gly Glu Asp Val Ser Met Asn Cys Thr Ser Ser Ser Ile Phe
355 360 365
Asn Thr Trp Leu Trp Tyr Lys Gln Glu Pro Gly Glu Gly Pro Val Leu
370 375 380
Leu Ile Ala Leu Tyr Lys Ala Gly Glu Leu Thr Ser Asn Gly Arg Leu
385 390 395 400
Thr Ala Gln Phe Gly Ile Thr Arg Lys Asp Ser Phe Leu Asn Ile Ser
405 410 415
Ala Ser Ile Pro Ser Asp Val Gly Ile Tyr Phe Cys Ala Gly Tyr Ser
420 425 430
Ser Ser Asn Asp Tyr Lys Leu Ser Phe Gly Ala Gly Thr Thr Val Thr
435 440 445
Val Arg Ala Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg
450 455 460
Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp
465 470 475 480
Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr
485 490 495
Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser
500 505 510
Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe
515 520 525
Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser
530 535 540
Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn
545 550 555 560
Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu
565 570 575
Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
580 585 590
<210> 416
<211> 638
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 40 полноразмерная последовательность, нативный
<400> 416
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ile Thr Ala Arg Ser Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser Leu
305 310 315 320
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met His Thr
325 330 335
Ser Thr Phe Gln Asn Arg Pro Gln Leu Phe Leu Leu Ile Trp Lys Lys
340 345 350
Leu Val Pro Gly Asn Pro Phe Arg Arg Ser Trp Met Lys Arg Glu Arg
355 360 365
Glu Met Leu Leu Ile Thr Ser Met Leu Val Leu Trp Met Gln Leu Ser
370 375 380
Gln Val Asn Gly Gln Gln Val Met Gln Ile Pro Gln Tyr Gln His Val
385 390 395 400
Gln Glu Gly Glu Asp Phe Thr Thr Tyr Cys Asn Ser Ser Thr Thr Leu
405 410 415
Ser Asn Ile Gln Trp Tyr Lys Gln Arg Pro Gly Gly His Pro Val Phe
420 425 430
Leu Ile Gln Leu Val Lys Ser Gly Glu Val Lys Lys Gln Lys Arg Leu
435 440 445
Thr Phe Gln Phe Gly Glu Ala Lys Lys Asn Ser Ser Leu His Ile Thr
450 455 460
Ala Thr Gln Thr Thr Asp Val Gly Thr Tyr Phe Cys Ala Gly Arg Asn
465 470 475 480
Asn Phe Asn Lys Phe Tyr Phe Gly Ser Gly Thr Lys Leu Asn Val Lys
485 490 495
Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser
500 505 510
Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln
515 520 525
Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys
530 535 540
Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val
545 550 555 560
Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn
565 570 575
Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys
580 585 590
Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn
595 600 605
Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val
610 615 620
Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
625 630 635
<210> 417
<211> 610
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 41 полноразмерная последовательность, нативный
<400> 417
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ser Asn Pro Arg Asp Arg Val Ser Tyr Glu Gln Tyr Phe Gly Pro Gly
115 120 125
Thr Arg Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu
130 135 140
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
145 150 155 160
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu
165 170 175
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr
180 185 190
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
195 200 205
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
210 215 220
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
225 230 235 240
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
245 250 255
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr
260 265 270
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
275 280 285
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
290 295 300
Met Val Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe
305 310 315 320
Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met
325 330 335
Met Ile Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu Ser
340 345 350
Trp Val Trp Ser Gln Arg Lys Glu Val Glu Gln Asp Pro Gly Pro Phe
355 360 365
Asn Val Pro Glu Gly Ala Thr Val Ala Phe Asn Cys Thr Tyr Ser Asn
370 375 380
Ser Ala Ser Gln Ser Phe Phe Trp Tyr Arg Gln Asp Cys Arg Lys Glu
385 390 395 400
Pro Lys Leu Leu Met Ser Val Tyr Ser Ser Gly Asn Glu Asp Gly Arg
405 410 415
Phe Thr Ala Gln Leu Asn Arg Ala Ser Gln Tyr Ile Ser Leu Leu Ile
420 425 430
Arg Asp Ser Lys Leu Ser Asp Ser Ala Thr Tyr Leu Cys Val Val Thr
435 440 445
Phe Ala Leu Thr Gly Gly Phe Lys Thr Ile Phe Gly Ala Gly Thr Arg
450 455 460
Leu Phe Val Lys Ala Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln
465 470 475 480
Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp
485 490 495
Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr
500 505 510
Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser
515 520 525
Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn
530 535 540
Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro
545 550 555 560
Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp
565 570 575
Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu
580 585 590
Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp
595 600 605
Ser Ser
610
<210> 418
<211> 638
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 42 полноразмерная последовательность, нативный
<400> 418
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Lys Thr Ser Arg Ser Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser Leu
305 310 315 320
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met His Thr
325 330 335
Ser Thr Phe Gln Asn Arg Pro Gln Leu Phe Leu Leu Ile Trp Lys Lys
340 345 350
Leu Val Pro Gly Asn Pro Phe Arg Arg Ser Trp Met Lys Arg Glu Arg
355 360 365
Glu Met Leu Leu Ile Thr Ser Met Leu Val Leu Trp Met Gln Leu Ser
370 375 380
Gln Val Asn Gly Gln Gln Val Met Gln Ile Pro Gln Tyr Gln His Val
385 390 395 400
Gln Glu Gly Glu Asp Phe Thr Thr Tyr Cys Asn Ser Ser Thr Thr Leu
405 410 415
Ser Asn Ile Gln Trp Tyr Lys Gln Arg Pro Gly Gly His Pro Val Phe
420 425 430
Leu Ile Gln Leu Val Lys Ser Gly Glu Val Lys Lys Gln Lys Arg Leu
435 440 445
Thr Phe Gln Phe Gly Glu Ala Lys Lys Asn Ser Ser Leu His Ile Thr
450 455 460
Ala Thr Gln Thr Thr Asp Val Gly Thr Tyr Phe Cys Ala Gly Pro Asp
465 470 475 480
Asn Phe Asn Lys Phe Tyr Phe Gly Ser Gly Thr Lys Leu Asn Val Lys
485 490 495
Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser
500 505 510
Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln
515 520 525
Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys
530 535 540
Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val
545 550 555 560
Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn
565 570 575
Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys
580 585 590
Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn
595 600 605
Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val
610 615 620
Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
625 630 635
<210> 419
<211> 609
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 43 полноразмерная последовательность, нативный
<400> 419
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ser Thr Pro Arg Asp Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser Leu
305 310 315 320
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Met Lys
325 330 335
Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu Ser Trp Val
340 345 350
Trp Ser Gln Gln Lys Glu Val Glu Gln Asn Ser Gly Pro Leu Ser Val
355 360 365
Pro Glu Gly Ala Ile Ala Ser Leu Asn Cys Thr Tyr Ser Asp Arg Gly
370 375 380
Ser Gln Ser Phe Phe Trp Tyr Arg Gln Tyr Ser Gly Lys Ser Pro Glu
385 390 395 400
Leu Ile Met Phe Ile Tyr Ser Asn Gly Asp Lys Glu Asp Gly Arg Phe
405 410 415
Thr Ala Gln Leu Asn Lys Ala Ser Gln Tyr Val Ser Leu Leu Ile Arg
420 425 430
Asp Ser Gln Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala Val Asn Val
435 440 445
Pro Thr Ser Gly Thr Tyr Lys Tyr Ile Phe Gly Thr Gly Thr Arg Leu
450 455 460
Lys Val Leu Ala Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu
465 470 475 480
Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe
485 490 495
Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile
500 505 510
Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn
515 520 525
Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala
530 535 540
Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu
545 550 555 560
Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr
565 570 575
Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu
580 585 590
Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser
595 600 605
Ser
<210> 420
<211> 616
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 44 полноразмерная последовательность, нативный
<400> 420
Met Gly Phe Arg Leu Leu Cys Cys Val Ala Phe Cys Leu Leu Gly Ala
1 5 10 15
Gly Pro Val Asp Ser Gly Val Thr Gln Thr Pro Lys His Leu Ile Thr
20 25 30
Ala Thr Gly Gln Arg Val Thr Leu Arg Cys Ser Pro Arg Ser Gly Asp
35 40 45
Leu Ser Val Tyr Trp Tyr Gln Gln Ser Leu Asp Gln Gly Leu Gln Phe
50 55 60
Leu Ile Gln Tyr Tyr Asn Gly Glu Glu Arg Ala Lys Gly Asn Ile Leu
65 70 75 80
Glu Arg Phe Ser Ala Gln Gln Phe Pro Asp Leu His Ser Glu Leu Asn
85 90 95
Leu Ser Ser Leu Glu Leu Gly Asp Ser Ala Leu Tyr Phe Cys Ala Ser
100 105 110
Ser Gly Thr Pro Asp Thr Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr
115 120 125
Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe
130 135 140
Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val
145 150 155 160
Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp
165 170 175
Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro
180 185 190
Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser
195 200 205
Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe
210 215 220
Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr
225 230 235 240
Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp
245 250 255
Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val
260 265 270
Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu
275 280 285
Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg
290 295 300
Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu Lys
305 310 315 320
Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Ala Gln Glu Leu
325 330 335
Gly Met Gln Cys Gln Ala Arg Gly Ile Leu Gln Gln Met Trp Gly Val
340 345 350
Phe Leu Leu Tyr Val Ser Met Lys Met Gly Gly Thr Thr Gly Gln Asn
355 360 365
Ile Asp Gln Pro Thr Glu Met Thr Ala Thr Glu Gly Ala Ile Val Gln
370 375 380
Ile Asn Cys Thr Tyr Gln Thr Ser Gly Phe Asn Gly Leu Phe Trp Tyr
385 390 395 400
Gln Gln His Ala Gly Glu Ala Pro Thr Phe Leu Ser Tyr Asn Val Leu
405 410 415
Asp Gly Leu Glu Glu Lys Gly Arg Phe Ser Ser Phe Leu Ser Arg Ser
420 425 430
Lys Gly Tyr Ser Tyr Leu Leu Leu Lys Glu Leu Gln Met Lys Asp Ser
435 440 445
Ala Ser Tyr Leu Cys Ala Gln Tyr Ser Gly Gly Tyr Gln Lys Val Thr
450 455 460
Phe Gly Thr Gly Thr Lys Leu Gln Val Ile Pro Asn Ile Gln Asn Pro
465 470 475 480
Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser
485 490 495
Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser
500 505 510
Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg
515 520 525
Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser
530 535 540
Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp
545 550 555 560
Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu
565 570 575
Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val
580 585 590
Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu
595 600 605
Met Thr Leu Arg Leu Trp Ser Ser
610 615
<210> 421
<211> 642
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 45 полноразмерная последовательность, нативный
<400> 421
Met Gly Thr Arg Leu Leu Phe Trp Val Ala Phe Cys Leu Leu Gly Ala
1 5 10 15
Tyr His Thr Gly Ala Gly Val Ser Gln Ser Pro Ser Asn Lys Val Thr
20 25 30
Glu Lys Gly Lys Asp Val Glu Leu Arg Cys Asp Pro Ile Ser Gly His
35 40 45
Thr Ala Leu Tyr Trp Tyr Arg Gln Arg Leu Gly Gln Gly Leu Glu Phe
50 55 60
Leu Ile Tyr Phe Gln Gly Asn Ser Ala Pro Asp Lys Ser Gly Leu Pro
65 70 75 80
Ser Asp Arg Phe Ser Ala Glu Arg Thr Gly Glu Ser Val Ser Thr Leu
85 90 95
Thr Ile Gln Arg Thr Gln Gln Glu Asp Ser Ala Val Tyr Leu Cys Ala
100 105 110
Ser Ser Leu Tyr Leu Gly Thr Thr Gly Glu Leu Phe Phe Gly Glu Gly
115 120 125
Ser Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu
130 135 140
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
145 150 155 160
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu
165 170 175
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr
180 185 190
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
195 200 205
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
210 215 220
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
225 230 235 240
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
245 250 255
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr
260 265 270
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
275 280 285
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
290 295 300
Met Val Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe
305 310 315 320
Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met
325 330 335
His Thr Ser Thr Phe Gln Asn Arg Pro Gln Leu Phe Leu Leu Ile Trp
340 345 350
Lys Lys Leu Val Pro Gly Asn Pro Phe Arg Arg Ser Trp Met Lys Arg
355 360 365
Glu Arg Glu Met Leu Leu Ile Thr Ser Met Leu Val Leu Trp Met Gln
370 375 380
Leu Ser Gln Val Asn Gly Gln Gln Val Met Gln Ile Pro Gln Tyr Gln
385 390 395 400
His Val Gln Glu Gly Glu Asp Phe Thr Thr Tyr Cys Asn Ser Ser Thr
405 410 415
Thr Leu Ser Asn Ile Gln Trp Tyr Lys Gln Arg Pro Gly Gly His Pro
420 425 430
Val Phe Leu Ile Gln Leu Val Lys Ser Gly Glu Val Lys Lys Gln Lys
435 440 445
Arg Leu Thr Phe Gln Phe Gly Glu Ala Lys Lys Asn Ser Ser Leu His
450 455 460
Ile Thr Ala Thr Gln Thr Thr Asp Val Gly Thr Tyr Phe Cys Ala Gly
465 470 475 480
Ser Ser Gly Ala Gly Ser Tyr Gln Leu Thr Phe Gly Lys Gly Thr Lys
485 490 495
Leu Ser Val Ile Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln
500 505 510
Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp
515 520 525
Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr
530 535 540
Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser
545 550 555 560
Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn
565 570 575
Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro
580 585 590
Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp
595 600 605
Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu
610 615 620
Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp
625 630 635 640
Ser Ser
<210> 422
<211> 609
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 46 полноразмерная последовательность, нативный
<400> 422
Met Gly Thr Arg Leu Leu Cys Trp Ala Ala Leu Cys Leu Leu Gly Ala
1 5 10 15
Asp His Thr Gly Ala Gly Val Ser Gln Thr Pro Ser Asn Lys Val Thr
20 25 30
Glu Lys Gly Lys Tyr Val Glu Leu Arg Cys Asp Pro Ile Ser Gly His
35 40 45
Thr Ala Leu Tyr Trp Tyr Arg Gln Ser Leu Gly Gln Gly Pro Glu Phe
50 55 60
Leu Ile Tyr Phe Gln Gly Thr Gly Ala Ala Asp Asp Ser Gly Leu Pro
65 70 75 80
Asn Asp Arg Phe Phe Ala Val Arg Pro Glu Gly Ser Val Ser Thr Leu
85 90 95
Lys Ile Gln Arg Thr Glu Arg Gly Asp Ser Ala Val Tyr Leu Cys Ala
100 105 110
Ser Ser Leu Tyr Leu Gly Gly Ser Glu Thr Gln Tyr Phe Gly Pro Gly
115 120 125
Thr Arg Leu Leu Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu
130 135 140
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
145 150 155 160
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu
165 170 175
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr
180 185 190
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
195 200 205
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
210 215 220
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
225 230 235 240
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
245 250 255
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr
260 265 270
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
275 280 285
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
290 295 300
Met Val Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe
305 310 315 320
Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met
325 330 335
Leu Leu Leu Leu Val Pro Ala Phe Gln Val Ile Phe Thr Leu Gly Gly
340 345 350
Thr Arg Ala Gln Ser Val Thr Gln Leu Asp Ser Gln Val Pro Val Phe
355 360 365
Glu Glu Ala Pro Val Glu Leu Arg Cys Asn Tyr Ser Ser Ser Val Ser
370 375 380
Val Tyr Leu Phe Trp Tyr Val Gln Tyr Pro Asn Gln Gly Leu Gln Leu
385 390 395 400
Leu Leu Lys Tyr Leu Ser Gly Ser Thr Leu Val Lys Gly Ile Asn Gly
405 410 415
Phe Glu Ala Glu Phe Asn Lys Ser Gln Thr Ser Phe His Leu Arg Lys
420 425 430
Pro Ser Val His Ile Ser Asp Thr Ala Glu Tyr Phe Cys Ala Val Ser
435 440 445
Pro Ser Ser Gly Thr Tyr Lys Tyr Ile Phe Gly Thr Gly Thr Arg Leu
450 455 460
Lys Val Leu Ala Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu
465 470 475 480
Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe
485 490 495
Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile
500 505 510
Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn
515 520 525
Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala
530 535 540
Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu
545 550 555 560
Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr
565 570 575
Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu
580 585 590
Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser
595 600 605
Ser
<210> 423
<211> 609
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 47 полноразмерная последовательность, нативный
<400> 423
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Met Thr Gly Arg Thr Thr Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser Leu
305 310 315 320
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Met Lys
325 330 335
Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu Ser Trp Val
340 345 350
Trp Ser Gln Gln Lys Glu Val Glu Gln Asn Ser Gly Pro Leu Ser Val
355 360 365
Pro Glu Gly Ala Ile Ala Ser Leu Asn Cys Thr Tyr Ser Asp Arg Gly
370 375 380
Ser Gln Ser Phe Phe Trp Tyr Arg Gln Tyr Ser Gly Lys Ser Pro Glu
385 390 395 400
Leu Ile Met Phe Ile Tyr Ser Asn Gly Asp Lys Glu Asp Gly Arg Phe
405 410 415
Thr Ala Gln Leu Asn Lys Ala Ser Gln Tyr Val Ser Leu Leu Ile Arg
420 425 430
Asp Ser Gln Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala Val Asn Leu
435 440 445
Leu Ser Gly Ser Ala Arg Gln Leu Thr Phe Gly Ser Gly Thr Gln Leu
450 455 460
Thr Val Leu Pro Asp Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu
465 470 475 480
Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe
485 490 495
Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile
500 505 510
Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn
515 520 525
Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala
530 535 540
Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu
545 550 555 560
Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr
565 570 575
Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu
580 585 590
Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser
595 600 605
Ser
<210> 424
<211> 607
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 48 полноразмерная последовательность, нативный
<400> 424
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ser Thr Gly Arg Val Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser Leu
305 310 315 320
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Met Lys
325 330 335
Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu Ser Trp Val
340 345 350
Trp Ser Gln Gln Lys Glu Val Glu Gln Asp Pro Gly Pro Leu Ser Val
355 360 365
Pro Glu Gly Ala Ile Val Ser Leu Asn Cys Thr Tyr Ser Asn Ser Ala
370 375 380
Phe Gln Tyr Phe Met Trp Tyr Arg Gln Tyr Ser Arg Lys Gly Pro Glu
385 390 395 400
Leu Leu Met Tyr Thr Tyr Ser Ser Gly Asn Lys Glu Asp Gly Arg Phe
405 410 415
Thr Ala Gln Val Asp Lys Ser Ser Lys Tyr Ile Ser Leu Phe Ile Arg
420 425 430
Asp Ser Gln Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala Met Arg Ile
435 440 445
Gln Gly Ala Gln Lys Leu Val Phe Gly Gln Gly Thr Arg Leu Thr Ile
450 455 460
Asn Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp
465 470 475 480
Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser
485 490 495
Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp
500 505 510
Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala
515 520 525
Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn
530 535 540
Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser
545 550 555 560
Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu
565 570 575
Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys
580 585 590
Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600 605
<210> 425
<211> 609
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 49 полноразмерная последовательность, нативный
<400> 425
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ser Thr Pro Arg Tyr Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser Leu
305 310 315 320
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Met Lys
325 330 335
Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu Ser Trp Val
340 345 350
Trp Ser Gln Gln Lys Glu Val Glu Gln Asn Ser Gly Pro Leu Ser Val
355 360 365
Pro Glu Gly Ala Ile Ala Ser Leu Asn Cys Thr Tyr Ser Asp Arg Gly
370 375 380
Ser Gln Ser Phe Phe Trp Tyr Arg Gln Tyr Ser Gly Lys Ser Pro Glu
385 390 395 400
Leu Ile Met Phe Ile Tyr Ser Asn Gly Asp Lys Glu Asp Gly Arg Phe
405 410 415
Thr Ala Gln Leu Asn Lys Ala Ser Gln Tyr Val Ser Leu Leu Ile Arg
420 425 430
Asp Ser Gln Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala Val Asn Ile
435 440 445
Gly Thr Ser Gly Thr Tyr Lys Tyr Ile Phe Gly Thr Gly Thr Arg Leu
450 455 460
Lys Val Leu Ala Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu
465 470 475 480
Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe
485 490 495
Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile
500 505 510
Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn
515 520 525
Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala
530 535 540
Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu
545 550 555 560
Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr
565 570 575
Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu
580 585 590
Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser
595 600 605
Ser
<210> 426
<211> 604
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 50 полноразмерная последовательность, нативный
<400> 426
Met Gly Phe Arg Leu Leu Cys Cys Val Ala Phe Cys Leu Leu Gly Ala
1 5 10 15
Gly Pro Val Asp Ser Gly Val Thr Gln Thr Pro Lys His Leu Ile Thr
20 25 30
Ala Thr Gly Gln Arg Val Thr Leu Arg Cys Ser Pro Arg Ser Gly Asp
35 40 45
Leu Ser Val Tyr Trp Tyr Gln Gln Ser Leu Asp Gln Gly Leu Gln Phe
50 55 60
Leu Ile His Tyr Tyr Asn Gly Glu Glu Arg Ala Lys Gly Asn Ile Leu
65 70 75 80
Glu Arg Phe Ser Ala Gln Gln Phe Pro Asp Leu His Ser Glu Leu Asn
85 90 95
Leu Ser Ser Leu Glu Leu Gly Asp Ser Ala Leu Tyr Phe Cys Ala Ser
100 105 110
Ser Ala Thr Arg Asp Ala Tyr Gly Tyr Thr Phe Gly Ser Gly Thr Arg
115 120 125
Leu Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Phe Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu Lys
305 310 315 320
Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Lys Thr Phe Ala
325 330 335
Gly Phe Ser Phe Leu Phe Leu Trp Leu Gln Leu Asp Cys Met Ser Arg
340 345 350
Gly Glu Asp Val Glu Gln Ser Leu Phe Leu Ser Val Arg Glu Gly Asp
355 360 365
Ser Ser Val Ile Asn Cys Thr Tyr Thr Asp Ser Ser Ser Thr Tyr Leu
370 375 380
Tyr Trp Tyr Lys Gln Glu Pro Gly Ala Gly Leu Gln Leu Leu Thr Tyr
385 390 395 400
Ile Phe Ser Asn Met Asp Met Lys Gln Asp Gln Arg Leu Thr Val Leu
405 410 415
Leu Asn Lys Lys Asp Lys His Leu Ser Leu Arg Ile Ala Asp Thr Gln
420 425 430
Thr Gly Asp Ser Ala Ile Tyr Phe Cys Ala Glu Ser Pro Pro Gly Thr
435 440 445
Tyr Lys Tyr Ile Phe Gly Thr Gly Thr Arg Leu Lys Val Leu Ala Asn
450 455 460
Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser
465 470 475 480
Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn
485 490 495
Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr Val
500 505 510
Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp
515 520 525
Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile
530 535 540
Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val
545 550 555 560
Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln
565 570 575
Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly
580 585 590
Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600
<210> 427
<211> 610
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 51 полноразмерная последовательность, нативный
<400> 427
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ile Ala Ser Arg Val Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser Leu
305 310 315 320
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Met Lys
325 330 335
Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu Ser Trp Val
340 345 350
Trp Ser Gln Gln Lys Glu Val Glu Gln Asn Ser Gly Pro Leu Ser Val
355 360 365
Pro Glu Gly Ala Ile Ala Ser Leu Asn Cys Thr Tyr Ser Asp Arg Gly
370 375 380
Ser Gln Ser Phe Phe Trp Tyr Arg Gln Tyr Ser Gly Lys Ser Pro Glu
385 390 395 400
Leu Ile Met Ser Ile Tyr Ser Asn Gly Asp Lys Glu Asp Gly Arg Phe
405 410 415
Thr Ala Gln Leu Asn Lys Ala Ser Gln Tyr Val Ser Leu Leu Ile Arg
420 425 430
Asp Ser Gln Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala Val Asn Met
435 440 445
Arg Gly Gly Gly Ser Asn Tyr Lys Leu Thr Phe Gly Lys Gly Thr Leu
450 455 460
Leu Thr Val Asn Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln
465 470 475 480
Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp
485 490 495
Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr
500 505 510
Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser
515 520 525
Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn
530 535 540
Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro
545 550 555 560
Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp
565 570 575
Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu
580 585 590
Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp
595 600 605
Ser Ser
610
<210> 428
<211> 601
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 52 полноразмерная последовательность, нативный
<400> 428
Met Gly Phe Arg Leu Leu Cys Cys Val Ala Phe Cys Leu Leu Gly Ala
1 5 10 15
Gly Pro Val Asp Ser Gly Val Thr Gln Thr Pro Lys His Leu Ile Thr
20 25 30
Ala Thr Gly Gln Arg Val Thr Leu Arg Cys Ser Pro Arg Ser Gly Asp
35 40 45
Leu Ser Val Tyr Trp Tyr Gln Gln Ser Leu Asp Gln Gly Leu Gln Phe
50 55 60
Leu Ile Gln Tyr Tyr Asn Gly Glu Glu Arg Ala Lys Gly Asn Ile Leu
65 70 75 80
Glu Arg Phe Ser Ala Gln Gln Phe Pro Asp Leu His Ser Glu Leu Asn
85 90 95
Leu Ser Ser Leu Glu Leu Gly Asp Ser Ala Leu Tyr Phe Cys Ala Ser
100 105 110
Ser Val Gly Asp Leu Asn Asn Glu Gln Phe Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser Leu
305 310 315 320
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Val Leu
325 330 335
Lys Phe Ser Val Ser Ile Leu Trp Ile Gln Leu Ala Trp Val Ser Thr
340 345 350
Gln Leu Leu Glu Gln Ser Pro Gln Phe Leu Ser Ile Gln Glu Gly Glu
355 360 365
Asn Leu Thr Val Tyr Cys Asn Ser Ser Ser Val Phe Ser Ser Leu Gln
370 375 380
Trp Tyr Arg Gln Glu Pro Gly Glu Gly Pro Val Leu Leu Val Thr Val
385 390 395 400
Val Thr Gly Gly Glu Val Lys Lys Leu Lys Arg Leu Thr Phe Gln Phe
405 410 415
Gly Asp Ala Arg Lys Asp Ser Ser Leu His Ile Thr Ala Ala Gln Pro
420 425 430
Gly Asp Thr Gly Leu Tyr Leu Cys Ala Gly Ala Arg Asp Tyr Lys Leu
435 440 445
Ser Phe Gly Ala Gly Thr Thr Val Thr Val Arg Ala Asn Ile Gln Asn
450 455 460
Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys
465 470 475 480
Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln
485 490 495
Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr Val Leu Asp Met
500 505 510
Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys
515 520 525
Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu
530 535 540
Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val
545 550 555 560
Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser
565 570 575
Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu
580 585 590
Leu Met Thr Leu Arg Leu Trp Ser Ser
595 600
<210> 429
<211> 611
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 53 полноразмерная последовательность, нативный
<400> 429
Met Gly Thr Ser Leu Leu Cys Trp Val Val Leu Gly Phe Leu Gly Thr
1 5 10 15
Asp His Thr Gly Ala Gly Val Ser Gln Ser Pro Arg Tyr Lys Val Thr
20 25 30
Lys Arg Gly Gln Asp Val Ala Leu Arg Cys Asp Pro Ile Ser Gly His
35 40 45
Val Ser Leu Tyr Trp Tyr Arg Gln Ala Leu Gly Gln Gly Pro Glu Phe
50 55 60
Leu Thr Tyr Phe Asn Tyr Glu Ala Gln Gln Asp Lys Ser Gly Leu Pro
65 70 75 80
Asn Asp Arg Phe Ser Ala Glu Arg Pro Glu Gly Ser Ile Ser Thr Leu
85 90 95
Thr Ile Gln Arg Thr Glu Gln Arg Asp Ser Ala Met Tyr Arg Cys Ala
100 105 110
Ser Ser Gly Ser Gly Thr Ser Gly Tyr Asn Glu Gln Phe Phe Gly Pro
115 120 125
Gly Thr Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro
130 135 140
Glu Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln
145 150 155 160
Lys Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val
165 170 175
Glu Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser
180 185 190
Thr Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg
195 200 205
Tyr Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn
210 215 220
Pro Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu
225 230 235 240
Asn Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val
245 250 255
Ser Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser
260 265 270
Tyr Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu
275 280 285
Gly Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met
290 295 300
Ala Met Val Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn
305 310 315 320
Phe Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro
325 330 335
Met Ala Ser Ala Pro Ile Ser Met Leu Ala Met Leu Phe Thr Leu Ser
340 345 350
Gly Leu Arg Ala Gln Ser Val Ala Gln Pro Glu Asp Gln Val Asn Val
355 360 365
Ala Glu Gly Asn Pro Leu Thr Val Lys Cys Thr Tyr Ser Val Ser Gly
370 375 380
Asn Pro Tyr Leu Phe Trp Tyr Val Gln Tyr Pro Asn Arg Gly Leu Gln
385 390 395 400
Phe Leu Leu Lys Tyr Ile Thr Gly Asp Asn Leu Val Lys Gly Ser Tyr
405 410 415
Gly Phe Glu Ala Glu Phe Asn Lys Ser Gln Thr Ser Phe His Leu Lys
420 425 430
Lys Pro Ser Ala Leu Val Ser Asp Ser Ala Leu Tyr Phe Cys Ala Val
435 440 445
Arg Asp Phe Gly Ser Gly Thr Tyr Lys Tyr Ile Phe Gly Thr Gly Thr
450 455 460
Arg Leu Lys Val Leu Ala Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr
465 470 475 480
Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr
485 490 495
Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val
500 505 510
Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys
515 520 525
Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala
530 535 540
Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser
545 550 555 560
Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr
565 570 575
Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile
580 585 590
Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu
595 600 605
Trp Ser Ser
610
<210> 430
<211> 472
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 16 - альфа нативный
<400> 430
agagtgttta tcttgtgagc cattctccat atttcagata taagatttca gttctcagtg 60
agtctaagtg acagaaggaa tggagaccct cttgggcctg cttatccttt ggctgcagct 120
gcaatgggtg agcagcaaac aggaggtgac gcagattcct gcagctctga gtgtcccaga 180
aggagaaaac ttggttctca actgcagttt cactgatagc gctatttaca acctccagtg 240
gtttaggcag gaccctggga aaggtctcac atctctgttg cttattcagt caagtcagag 300
agagcaaaca agtggaagac ttaatgcctc gctggataaa tcatcaggac gtagtacttt 360
atacattgca gcttctcagc ctggtgactc agccacctac ctctgtgctg tgaggcccgc 420
caataacaat gacatgcgct ttggagcagg gaccagactg acagtaaaac ca 472
<210> 431
<211> 420
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 16 - бета нативный
<400> 431
gattctttct tcaaagcagc catgggaatc aggctcctct gtcgtgtggc cttttgtttc 60
ctggctgtag gcctcgtaga tgtgaaagta acccagagct cgagatatct agtcaaaagg 120
acgggagaga aagtttttct ggaatgtgtc caggatatgg accatgaaaa tatgttctgg 180
tatcgacaag acccaggtct ggggctacgg ctgatctatt tctcatatga tgttaaaatg 240
aaagaaaaag gagatattcc tgaggggtac agtgtctcta gagagaagaa ggagcgcttc 300
tccctgattc tggagtccgc cagcaccaac cagacatcta tgtacctctg tgccagcagt 360
ttatgggggc gcagcaatca gccccagcat tttggtgatg ggactcgact ctccatccta 420
<210> 432
<211> 1833
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 15 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 432
atggacagct ggaccttttg ctgcgtgtcc ctgtgcatcc tggtggccaa gcacaccgat 60
gccggcgtga tccagagccc cagacacgaa gtgaccgaga tgggccagga agtgaccctg 120
cgctgcaagc ccatcagcgg ccacaacagc ctgttctggt acagacagac catgatgcgg 180
ggcctggaac tgctgatcta cttcaacaac aacgtgccca tcgacgacag cggcatgccc 240
gaggacagat tcagcgccaa gatgcccaac gccagcttca gcaccctgaa gatccagccc 300
agcgagccca gagacagcgc cgtgtacttt tgcgccagca gcctcgtggg cagaagccgg 360
accgagcagt actttggccc tggcaccaga ctgaccgtga ccgaagatct gaagaacgtc 420
ttcccccctg aggtggcagt ctttgaaccc tctgaggccg aaatcagtca cactcagaaa 480
gccaccctgg tgtgcctggc tacagggttt taccctgatc acgtggagct gagttggtgg 540
gtcaacggca aggaagtgca ttcaggggtc tgcactgacc cacagcccct gaaagagcag 600
ccagcactga atgattccag gtactgtctg agttcacggc tgagagtgtc tgccacattt 660
tggcagaacc cccgaaatca tttccggtgc caggtccagt tttatggcct gagcgagaat 720
gacgaatgga cacaggatcg ggctaagccc gtgactcaga ttgtctccgc cgaggcttgg 780
gggagagcag actgtggatt cacctccgaa tcttaccagc agggcgtgct gtctgccacc 840
atcctgtacg agattctgct ggggaaggct acactgtatg ccgtgctggt gagcgccctg 900
gtgctgatgg caatggtcaa gagaaaagac agcaggggag gctctggcgc caccaacttt 960
agcctgctga agcaggccgg cgatgtggag gagaatcctg gcccaatgag cctgagcagc 1020
ctgctgaagg tggtcaccgc ttctctgtgg ctgggccctg gaatcgccca gaagatcacc 1080
cagacccagc ccggcatgtt cgtgcaggaa aaagaggccg tcaccctgga ctgcacctac 1140
gacaccagcg accagagcta cggcctgttc tggtacaagc agcccagcag cggcgagatg 1200
atcttcctga tctaccaggg cagctacgac gagcagaacg ccaccgaggg ccggtacagc 1260
ctgaacttcc agaaggcccg gaagtccgcc aacctggtca tctctgccag ccagctgggc 1320
gacagcgcca tgtacttttg cgccatgaag cctggcggct acaacaagct gatcttcgga 1380
gccggcacca gactggccgt gcacccttat atccagaatc ccgaccctgc cgtctaccag 1440
ctgagggact caaagagctc cgataaaagc gtgtgcctgt tcaccgactt tgattctcag 1500
acaaacgtca gccagtccaa ggacagtgac gtgtacatta cagacaaatg cgtgctggat 1560
atgcgctcca tggacttcaa gtctaacagt gcagtggcct ggtccaacaa gtctgatttc 1620
gcttgcgcaa acgcctttaa caattcaatc attccagagg ataccttctt tccaagcccc 1680
gaatctagtt gtgacgtgaa gctggtcgag aaatctttcg aaacagatac taacctgaat 1740
tttcagaatc tgagcgtgat cggcttccgc attctgctgc tgaaagtggc tgggtttaat 1800
ctgctgatga ctctgcggct gtggtcttcc taa 1833
<210> 433
<211> 1815
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 16 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 433
atgggcatca gactgctgtg cagagtggcc ttctgcttcc tggccgtggg cctggtggac 60
gtgaaagtga cccagagcag cagatacctg gtcaagcgga ccggcgagaa ggtgttcctg 120
gaatgcgtgc aggacatgga ccacgagaat atgttctggt acagacagga ccccggcctg 180
ggcctgcggc tgatctactt cagctacgac gtgaagatga aggaaaaggg cgacatcccc 240
gagggctaca gcgtgtccag agagaagaaa gagcggttca gcctgatcct ggaaagcgcc 300
agcaccaacc agaccagcat gtacctgtgc gccagcagcc tgtggggcag aagcaaccag 360
cctcagcact tcggcgacgg caccagactg agcatcctcg aggacctgaa taaggtgttc 420
ccccctgagg tggccgtgtt tgagccaagc gaggccgaga tctcccacac ccagaaggcc 480
accctggtgt gcctggcaac cggcttcttt cccgatcacg tggagctgtc ctggtgggtg 540
aacggcaagg aggtgcactc tggcgtgtgc acagacccac agcccctgaa ggagcagcct 600
gccctgaatg attcccgcta ttgtctgtcc tctcggctga gagtgtctgc caccttttgg 660
cagaacccac ggaatcactt cagatgccag gtgcagtttt acggcctgtc tgagaacgac 720
gagtggaccc aggatcgggc caagcctgtg acacagatcg tgagcgcgga agcatggggc 780
agagccgact gtggcttcac cagcgtgtcc tatcagcagg gcgtgctgtc cgccaccatc 840
ctgtacgaga tcctgctggg caaggccaca ctgtatgccg tgctggtgtc tgccctggtg 900
ctgatggcca tggtgaagag aaaagacttc ggctctggcg ccaccaactt tagcctgctg 960
aagcaggccg gcgatgtgga ggagaatcct ggcccaatgg aaaccctgct gggcctgctg 1020
atcctgtggc tgcagctcca gtgggtgtcc agcaagcagg aagtgaccca gatccctgcc 1080
gccctgtctg tgcctgaggg cgagaacctg gtgctgaact gcagcttcac cgacagcgcc 1140
atctacaacc tgcagtggtt ccggcaggac cccggcaagg gactgacaag cctgctgctg 1200
attcagagca gccagagaga gcagaccagc ggcagactga acgccagcct ggacaagtcc 1260
tccggcagaa gcaccctgta tatcgccgcc tcccagcctg gcgatagcgc cacatatctg 1320
tgtgccgtgc ggcctgccaa caacaacgac atgagattcg gagccggcac ccggctgacc 1380
gtgaagccta atatccagaa tcccgaccct gccgtctacc agctgaggga ctcaaagagc 1440
tccgataaaa gcgtgtgcct gttcaccgac tttgattctc agacaaacgt cagccagtcc 1500
aaggacagtg acgtgtacat tacagacaaa tgcgtgctgg atatgcgctc catggacttc 1560
aagtctaaca gtgcagtggc ctggtccaac aagtctgatt tcgcttgcgc aaacgccttt 1620
aacaattcaa tcattccaga ggataccttc tttccaagcc ccgaatctag ttgtgacgtg 1680
aagctggtcg agaaatcttt cgaaacagat actaacctga attttcagaa tctgagcgtg 1740
atcggcttcc gcattctgct gctgaaagtg gctgggttta atctgctgat gactctgcgg 1800
ctgtggtctt cctaa 1815
<210> 434
<211> 1824
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 17 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 434
atgggcatca gactgctgtg cagagtggcc ttctgcttcc tggccgtggg cctggtggac 60
gtgaaagtga cccagagcag cagatacctg gtcaagcgga ccggcgagaa ggtgttcctg 120
gaatgcgtgc aggacatgga ccacgagaat atgttctggt acagacagga ccccggcctg 180
ggcctgcggc tgatctactt cagctacgac gtgaagatga aggaaaaggg cgacatcccc 240
gagggctaca gcgtgtccag agagaagaaa gagcggttca gcctgatcct ggaaagcgcc 300
agcaccaacc agaccagcat gtacctgtgc gccagcagcc tgtggggcag aagcaaccag 360
cctcagcact tcggcgacgg caccagactg agcatcctcg aggacctgaa taaggtgttc 420
ccccctgagg tggccgtgtt tgagccaagc gaggccgaga tctcccacac ccagaaggcc 480
accctggtgt gcctggcaac cggcttcttt cccgatcacg tggagctgtc ctggtgggtg 540
aacggcaagg aggtgcactc tggcgtgtgc acagacccac agcccctgaa ggagcagcct 600
gccctgaatg attcccgcta ttgtctgtcc tctcggctga gagtgtctgc caccttttgg 660
cagaacccac ggaatcactt cagatgccag gtgcagtttt acggcctgtc tgagaacgac 720
gagtggaccc aggatcgggc caagcctgtg acacagatcg tgagcgcgga agcatggggc 780
agagccgact gtggcttcac cagcgtgtcc tatcagcagg gcgtgctgtc cgccaccatc 840
ctgtacgaga tcctgctggg caaggccaca ctgtatgccg tgctggtgtc tgccctggtg 900
ctgatggcca tggtgaagag aaaagacttc ggctctggcg ccaccaactt tagcctgctg 960
aagcaggccg gcgatgtgga ggagaatcct ggcccaatga gcctgagcag cctgctgaag 1020
gtggtcaccg cttctctgtg gctgggccct ggaatcgccc agaagatcac ccagacccag 1080
cccggcatgt tcgtgcagga aaaagaggcc gtcaccctgg actgcaccta cgacaccagc 1140
gaccccagct acggcctgtt ctggtacaag cagcccagca gcggcgagat gatcttcctg 1200
atctaccagg gcagctacga ccagcagaac gccaccgagg gccggtacag cctgaacttc 1260
cagaaggccc ggaagtccgc caacctggtc atctctgcca gccagctggg cgacagcgcc 1320
atgtactttt gcgccatgag agagggcaga ggcgacaaga tcattttcgg caagggcacc 1380
cggctgcaca tcctgcccaa tatccagaat cccgaccctg ccgtctacca gctgagggac 1440
tcaaagagct ccgataaaag cgtgtgcctg ttcaccgact ttgattctca gacaaacgtc 1500
agccagtcca aggacagtga cgtgtacatt acagacaaat gcgtgctgga tatgcgctcc 1560
atggacttca agtctaacag tgcagtggcc tggtccaaca agtctgattt cgcttgcgca 1620
aacgccttta acaattcaat cattccagag gataccttct ttccaagccc cgaatctagt 1680
tgtgacgtga agctggtcga gaaatctttc gaaacagata ctaacctgaa ttttcagaat 1740
ctgagcgtga tcggcttccg cattctgctg ctgaaagtgg ctgggtttaa tctgctgatg 1800
actctgcggc tgtggtcttc ctaa 1824
<210> 435
<211> 1824
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 18 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 435
atgggcatca gactgctgtg cagagtggcc ttctgcttcc tggccgtggg cctggtggac 60
gtgaaagtga cccagagcag cagatacctg gtcaagcgga ccggcgagaa ggtgttcctg 120
gaatgcgtgc aggacatgga ccacgagaat atgttctggt acagacagga ccccggcctg 180
ggcctgcggc tgatctactt cagctacgac gtgaagatga aggaaaaggg cgacatcccc 240
gagggctaca gcgtgtccag agagaagaaa gagcggttca gcctgatcct ggaaagcgcc 300
agcaccaacc agaccagcat gtacctgtgc gccagcagct tctggggcag aagcaacagc 360
cctctgcact tcggcaacgg caccagactg accgtgaccg aggacctgaa taaggtgttc 420
ccccctgagg tggccgtgtt tgagccaagc gaggccgaga tctcccacac ccagaaggcc 480
accctggtgt gcctggcaac cggcttcttt cccgatcacg tggagctgtc ctggtgggtg 540
aacggcaagg aggtgcactc tggcgtgtgc acagacccac agcccctgaa ggagcagcct 600
gccctgaatg attcccgcta ttgtctgtcc tctcggctga gagtgtctgc caccttttgg 660
cagaacccac ggaatcactt cagatgccag gtgcagtttt acggcctgtc tgagaacgac 720
gagtggaccc aggatcgggc caagcctgtg acacagatcg tgagcgcgga agcatggggc 780
agagccgact gtggcttcac cagcgtgtcc tatcagcagg gcgtgctgtc cgccaccatc 840
ctgtacgaga tcctgctggg caaggccaca ctgtatgccg tgctggtgtc tgccctggtg 900
ctgatggcca tggtgaagag aaaagacttc ggctctggcg ccaccaactt tagcctgctg 960
aagcaggccg gcgatgtgga ggagaatcct ggcccaatgg aaaccctgct gggcctgctg 1020
atcctgtggc tgcagctcca gtgggtgtcc agcaagcagg aagtgaccca gatccctgcc 1080
gccctgtctg tgcctgaggg cgagaacctg gtgctgaact gcagcttcac cgacagcgcc 1140
atctacaacc tgcagtggtt ccggcaggac cccggcaagg gactgacaag cctgctgctg 1200
attcagagca gccagagaga gcagaccagc ggcagactga acgccagcct ggacaagtcc 1260
tccggcagaa gcaccctgta tatcgccgcc tcccagcctg gcgatagcgc cacatatctg 1320
tgcgccgagg gcaatgccgg cggaaccagc tacggcaagc tgacctttgg ccagggcacc 1380
atcctgaccg tgcaccccaa tatccagaat cccgaccctg ccgtctacca gctgagggac 1440
tcaaagagct ccgataaaag cgtgtgcctg ttcaccgact ttgattctca gacaaacgtc 1500
agccagtcca aggacagtga cgtgtacatt acagacaaat gcgtgctgga tatgcgctcc 1560
atggacttca agtctaacag tgcagtggcc tggtccaaca agtctgattt cgcttgcgca 1620
aacgccttta acaattcaat cattccagag gataccttct ttccaagccc cgaatctagt 1680
tgtgacgtga agctggtcga gaaatctttc gaaacagata ctaacctgaa ttttcagaat 1740
ctgagcgtga tcggcttccg cattctgctg ctgaaagtgg ctgggtttaa tctgctgatg 1800
actctgcggc tgtggtcttc ctaa 1824
<210> 436
<211> 1836
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 19 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 436
atgggcatca gactgctgtg cagagtggcc ttctgcttcc tggccgtggg cctggtggac 60
gtgaaagtga cccagagcag cagatacctg gtcaagcgga ccggcgagaa ggtgttcctg 120
gaatgcgtgc aggacatgga ccacgagaat atgttctggt acagacagga ccccggcctg 180
ggcctgcggc tgatctactt cagctacgac gtgaagatga aggaaaaggg cgacatcccc 240
gagggctaca gcgtgtccag agagaagaaa gagcggttca gcctgatcct ggaaagcgcc 300
agcaccaacc agaccagcat gtacctgtgc gccagcagca gctggggcca gagcacaggc 360
gagcagtact ttggccctgg caccagactg accgtgaccg aagatctgaa gaacgtcttc 420
ccccctgagg tggcagtctt tgaaccctct gaggccgaaa tcagtcacac tcagaaagcc 480
accctggtgt gcctggctac agggttttac cctgatcacg tggagctgag ttggtgggtc 540
aacggcaagg aagtgcattc aggggtctgc actgacccac agcccctgaa agagcagcca 600
gcactgaatg attccaggta ctgtctgagt tcacggctga gagtgtctgc cacattttgg 660
cagaaccccc gaaatcattt ccggtgccag gtccagtttt atggcctgag cgagaatgac 720
gaatggacac aggatcgggc taagcccgtg actcagattg tctccgccga ggcttggggg 780
agagcagact gtggattcac ctccgaatct taccagcagg gcgtgctgtc tgccaccatc 840
ctgtacgaga ttctgctggg gaaggctaca ctgtatgccg tgctggtgag cgccctggtg 900
ctgatggcaa tggtcaagag aaaagacagc aggggaggct ctggcgccac caactttagc 960
ctgctgaagc aggccggcga tgtggaggag aatcctggcc caatgagcct gagcagcctg 1020
ctgaaggtgg tcaccgcttc tctgtggctg ggccctggaa tcgcccagaa gatcacccag 1080
acccagcccg gcatgttcgt gcaggaaaaa gaggccgtca ccctggactg cacctacgac 1140
accagcgacc agagctacgg cctgttctgg tacaagcagc ccagcagcgg cgagatgatc 1200
ttcctgatct accagggcag ctacgacgag cagaacgcca ccgagggccg gtacagcctg 1260
aacttccaga aggcccggaa gtccgccaac ctggtcatct ctgccagcca gctgggcgac 1320
agcgccatgt acttttgcgc catgagagag aacaccggca ccgccagcaa gctgaccttc 1380
ggcacaggca ccagactgca ggtcacactg gacatccaga atcccgaccc tgccgtctac 1440
cagctgaggg actcaaagag ctccgataaa agcgtgtgcc tgttcaccga ctttgattct 1500
cagacaaacg tcagccagtc caaggacagt gacgtgtaca ttacagacaa atgcgtgctg 1560
gatatgcgct ccatggactt caagtctaac agtgcagtgg cctggtccaa caagtctgat 1620
ttcgcttgcg caaacgcctt taacaattca atcattccag aggatacctt ctttccaagc 1680
cccgaatcta gttgtgacgt gaagctggtc gagaaatctt tcgaaacaga tactaacctg 1740
aattttcaga atctgagcgt gatcggcttc cgcattctgc tgctgaaagt ggctgggttt 1800
aatctgctga tgactctgcg gctgtggtct tcctaa 1836
<210> 437
<211> 1827
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 20 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 437
atgctgctgc tcctgctcct cctgggacct ggatctggac tgggagccgt ggtgtcccag 60
caccccagca gagtgatctg caagagcggc accagcgtga agatcgagtg cagaagcctg 120
gacttccagg ccaccaccat gttctggtac agacagttcc ccaagcagag cctgatgctg 180
atggccacct ccaacgaggg cagcaaggcc acatacgagc agggcgtgga aaaggacaag 240
ttcctgatca accacgccag cctgaccctg agcaccctga cagtgacaag cgcccacccc 300
gaggacagca gcttctacat ctgtagcgcc agcagcctgg ccagaagaag ctacgagcag 360
tacttcggcc ctggcaccag actgaccgtg accgaagatc tgaagaacgt cttcccccct 420
gaggtggcag tctttgaacc ctctgaggcc gaaatcagtc acactcagaa agccaccctg 480
gtgtgcctgg ctacagggtt ttaccctgat cacgtggagc tgagttggtg ggtcaacggc 540
aaggaagtgc attcaggggt ctgcactgac ccacagcccc tgaaagagca gccagcactg 600
aatgattcca ggtactgtct gagttcacgg ctgagagtgt ctgccacatt ttggcagaac 660
ccccgaaatc atttccggtg ccaggtccag ttttatggcc tgagcgagaa tgacgaatgg 720
acacaggatc gggctaagcc cgtgactcag attgtctccg ccgaggcttg ggggagagca 780
gactgtggat tcacctccga atcttaccag cagggcgtgc tgtctgccac catcctgtac 840
gagattctgc tggggaaggc tacactgtat gccgtgctgg tgagcgccct ggtgctgatg 900
gcaatggtca agagaaaaga cagcagggga ggctctggcg ccaccaactt tagcctgctg 960
aagcaggccg gcgatgtgga ggagaatcct ggcccaatga acatgctgac cgccagcctg 1020
ctgagagccg tgatcgccag catctgcgtg gtgtccagca tggcccagaa agtgacccag 1080
gcccagaccg agatcagcgt ggtggaaaaa gaagatgtga ccctggactg cgtgtacgag 1140
acacgggaca ccacctacta cctgttctgg tacaagcagc ctcccagcgg cgagctggtg 1200
ttcctgatca gacggaacag cttcgacgag cagaacgaga ttagcggccg gtacagctgg 1260
aacttccaga agtccaccag cagcttcaac ttcaccatca ccgcctccca ggtggtggac 1320
agcgccgtgt acttttgtgc cctgtggacc ggggccaaca acctgttctt tggcaccgga 1380
acccggctga ccgtgatccc ctatatccag aatcccgacc ctgccgtcta ccagctgagg 1440
gactcaaaga gctccgataa aagcgtgtgc ctgttcaccg actttgattc tcagacaaac 1500
gtcagccagt ccaaggacag tgacgtgtac attacagaca aatgcgtgct ggatatgcgc 1560
tccatggact tcaagtctaa cagtgcagtg gcctggtcca acaagtctga tttcgcttgc 1620
gcaaacgcct ttaacaattc aatcattcca gaggatacct tctttccaag ccccgaatct 1680
agttgtgacg tgaagctggt cgagaaatct ttcgaaacag atactaacct gaattttcag 1740
aatctgagcg tgatcggctt ccgcattctg ctgctgaaag tggctgggtt taatctgctg 1800
atgactctgc ggctgtggtc ttcctaa 1827
<210> 438
<211> 1878
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 21 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 438
atgcacagac ctagaaggcc cctgcatccc gtggcccctg ccatgtctat cggcctgctg 60
tgctgcgtgg cattctccct gctgtgggcc tctcctgtga atgccggcgt gacccagacc 120
cctaagttcc aggtgctgaa aaccggccag agcatgaccc tgcagtgcgc ccaggacatg 180
aaccacaaca gcatgtactg gtacagacag gaccccggca tgggcctgcg gctgatctac 240
tatagcgcca gcgagggcac caccgacaag ggcgaggtgc ccaacggcta caacgtgtcc 300
cggctgaaca agagagagtt cagcctgaga ctggaaagcg ccgctcccag ccagaccagc 360
gtgtacttct gtgccagcag accctggggc aaccagaaca ccgaggcatt ctttgggcag 420
ggcacccggc tgacagtggt cgaggacctg aataaggtgt tcccccctga ggtggccgtg 480
tttgagccaa gcgaggccga gatctcccac acccagaagg ccaccctggt gtgcctggca 540
accggcttct ttcccgatca cgtggagctg tcctggtggg tgaacggcaa ggaggtgcac 600
tctggcgtgt gcacagaccc acagcccctg aaggagcagc ctgccctgaa tgattcccgc 660
tattgtctgt cctctcggct gagagtgtct gccacctttt ggcagaaccc acggaatcac 720
ttcagatgcc aggtgcagtt ttacggcctg tctgagaacg acgagtggac ccaggatcgg 780
gccaagcctg tgacacagat cgtgagcgcg gaagcatggg gcagagccga ctgtggcttc 840
accagcgtgt cctatcagca gggcgtgctg tccgccacca tcctgtacga gatcctgctg 900
ggcaaggcca cactgtatgc cgtgctggtg tctgccctgg tgctgatggc catggtgaag 960
agaaaagact tcggctctgg cgccaccaac tttagcctgc tgaagcaggc cggcgatgtg 1020
gaggagaatc ctggcccaat gagcctgagc agcctgctga aggtggtcac cgcttctctg 1080
tggctgggcc ctggaatcgc ccagaagatc acccagaccc agcccggcat gttcgtgcag 1140
gaaaaagagg ccgtcaccct ggactgcacc tacgacacca gcgaccagag ctacggcctg 1200
ttctggtaca agcagcccag cagcggcgag atgatcttcc tgatctacca gggcagctac 1260
gacgagcaga acgccaccga gggccggtac agcctgaact tccagaaggc ccggaagtcc 1320
gccaacctgg tcatctctgc cagccagctg ggcgacagcg ccatgtactt ttgcgccatg 1380
cgcgagggca gagtgaccgg cggaggcaac aagctgacct ttggcaccgg cacccagctg 1440
aaagtggaac tgaacatcca gaatcccgac cctgccgtct accagctgag ggactcaaag 1500
agctccgata aaagcgtgtg cctgttcacc gactttgatt ctcagacaaa cgtcagccag 1560
tccaaggaca gtgacgtgta cattacagac aaatgcgtgc tggatatgcg ctccatggac 1620
ttcaagtcta acagtgcagt ggcctggtcc aacaagtctg atttcgcttg cgcaaacgcc 1680
tttaacaatt caatcattcc agaggatacc ttctttccaa gccccgaatc tagttgtgac 1740
gtgaagctgg tcgagaaatc tttcgaaaca gatactaacc tgaattttca gaatctgagc 1800
gtgatcggct tccgcattct gctgctgaaa gtggctgggt ttaatctgct gatgactctg 1860
cggctgtggt cttcctaa 1878
<210> 439
<211> 1854
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 22 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 439
atgggacctg gactgctgtg ctgggtgctg ctgtgtctgc tgggagccgg acctgtggat 60
gccggcgtga cacagagccc cacccacctg atcaagacca gaggccagca cgtgaccctg 120
cggtgtagcc ctatcagcgg ccacaagagc gtgtcctggt atcagcaggt cctgggccag 180
ggccctcagt tcatcttcca gtactacgag aaagaggaac gcggcagagg caacttcccc 240
gacagattca gcgccagaca gttccccaac tacagcagcg agctgaacgt gaacgccctg 300
ctgctgggcg atagcgccct gtacctgtgt gccagcagcc ggaccgagaa ctacggctac 360
accttcggca gcggcaccag actgacagtg gtcgaggacc tgaataaggt gttcccccct 420
gaggtggccg tgtttgagcc aagcgaggcc gagatctccc acacccagaa ggccaccctg 480
gtgtgcctgg caaccggctt ctttcccgat cacgtggagc tgtcctggtg ggtgaacggc 540
aaggaggtgc actctggcgt gtgcacagac ccacagcccc tgaaggagca gcctgccctg 600
aatgattccc gctattgtct gtcctctcgg ctgagagtgt ctgccacctt ttggcagaac 660
ccacggaatc acttcagatg ccaggtgcag ttttacggcc tgtctgagaa cgacgagtgg 720
acccaggatc gggccaagcc tgtgacacag atcgtgagcg cggaagcatg gggcagagcc 780
gactgtggct tcaccagcgt gtcctatcag cagggcgtgc tgtccgccac catcctgtac 840
gagatcctgc tgggcaaggc cacactgtat gccgtgctgg tgtctgccct ggtgctgatg 900
gccatggtga agagaaaaga cttcggctct ggcgccacca actttagcct gctgaagcag 960
gccggcgatg tggaggagaa tcctggccca atggcccagg aactgggcat gcagtgtcag 1020
gccagaggca tcctgcagca gatgtggggc gtgttcctgc tgtacgtgtc catgaagatg 1080
ggcggcacca ccggccagaa catcgaccag cctaccgaga tgaccgccac cgagggcgct 1140
atcgtgcaga tcaactgcac ctaccagacc agcggcttca acggcctgtt ctggtatcag 1200
cagcacgccg gcgaggcccc taccttcctg agctacaatg tgctggacgg cctggaagag 1260
aagggccggt tcagcagctt cctgtccaga agcaagggct acagctacct gctgctgaaa 1320
gaactgcaga tgaaggacag cgcctcctac ctgtgtgccg tgcgggccag aatggacagc 1380
aactaccagc tgatctgggg agccggcacc aagctgatca tcaagcccga catccagaat 1440
cccgaccctg ccgtctacca gctgagggac tcaaagagct ccgataaaag cgtgtgcctg 1500
ttcaccgact ttgattctca gacaaacgtc agccagtcca aggacagtga cgtgtacatt 1560
acagacaaat gcgtgctgga tatgcgctcc atggacttca agtctaacag tgcagtggcc 1620
tggtccaaca agtctgattt cgcttgcgca aacgccttta acaattcaat cattccagag 1680
gataccttct ttccaagccc cgaatctagt tgtgacgtga agctggtcga gaaatctttc 1740
gaaacagata ctaacctgaa ttttcagaat ctgagcgtga tcggcttccg cattctgctg 1800
ctgaaagtgg ctgggtttaa tctgctgatg actctgcggc tgtggtcttc ctaa 1854
<210> 440
<211> 1830
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 23 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 440
atgggcatca gactgctgtg cagagtggcc ttctgcttcc tggccgtggg cctggtggac 60
gtgaaagtga cccagagcag cagatacctg gtcaagcgga ccggcgagaa ggtgttcctg 120
gaatgcgtgc aggacatgga ccacgagaat atgttctggt acagacagga ccccggcctg 180
ggcctgcggc tgatctactt cagctacgac gtgaagatga aggaaaaggg cgacatcccc 240
gagggctaca gcgtgtccag agagaagaaa gagcggttca gcctgatcct ggaaagcgcc 300
agcaccaacc agaccagcat gtacctgtgc gccagcagcc cctggggcca gtctaatcag 360
cctcagcact tcggcgacgg cacccggctg tctattctcg aggacctgaa taaggtgttc 420
ccccctgagg tggccgtgtt tgagccaagc gaggccgaga tctcccacac ccagaaggcc 480
accctggtgt gcctggcaac cggcttcttt cccgatcacg tggagctgtc ctggtgggtg 540
aacggcaagg aggtgcactc tggcgtgtgc acagacccac agcccctgaa ggagcagcct 600
gccctgaatg attcccgcta ttgtctgtcc tctcggctga gagtgtctgc caccttttgg 660
cagaacccac ggaatcactt cagatgccag gtgcagtttt acggcctgtc tgagaacgac 720
gagtggaccc aggatcgggc caagcctgtg acacagatcg tgagcgcgga agcatggggc 780
agagccgact gtggcttcac cagcgtgtcc tatcagcagg gcgtgctgtc cgccaccatc 840
ctgtacgaga tcctgctggg caaggccaca ctgtatgccg tgctggtgtc tgccctggtg 900
ctgatggcca tggtgaagag aaaagacttc ggctctggcg ccaccaactt tagcctgctg 960
aagcaggccg gcgatgtgga ggagaatcct ggcccaatga gcctgagcag cctgctgaag 1020
gtggtcaccg cttctctgtg gctgggccct ggaatcgccc agaagatcac ccagacccag 1080
cccggcatgt tcgtgcagga aaaagaggcc gtcaccctgg actgcaccta cgacaccagc 1140
gaccagagct acggcctgtt ctggtacaag cagcccagca gcggcgagat gatcttcctg 1200
atctaccagg gcagctacga cgagcagaac gccaccgagg gccggtacag cctgaacttc 1260
cagaaggccc ggaagtccgc caacctggtc atctctgcca gccagctggg cgacagcgcc 1320
atgtactttt gcgccatgag cccacctggc ggcagcgcca gacagctgac atttggctct 1380
ggcacccagc tgaccgtgct gcccgatatc cagaatcccg accctgccgt ctaccagctg 1440
agggactcaa agagctccga taaaagcgtg tgcctgttca ccgactttga ttctcagaca 1500
aacgtcagcc agtccaagga cagtgacgtg tacattacag acaaatgcgt gctggatatg 1560
cgctccatgg acttcaagtc taacagtgca gtggcctggt ccaacaagtc tgatttcgct 1620
tgcgcaaacg cctttaacaa ttcaatcatt ccagaggata ccttctttcc aagccccgaa 1680
tctagttgtg acgtgaagct ggtcgagaaa tctttcgaaa cagatactaa cctgaatttt 1740
cagaatctga gcgtgatcgg cttccgcatt ctgctgctga aagtggctgg gtttaatctg 1800
ctgatgactc tgcggctgtg gtcttcctaa 1830
<210> 441
<211> 1839
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 24 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 441
atgggcatca gactgctgtg cagagtggcc ttctgcttcc tggccgtggg cctggtggac 60
gtgaaagtga cccagagcag cagatacctg gtcaagcgga ccggcgagaa ggtgttcctg 120
gaatgcgtgc aggacatgga ccacgagaat atgttctggt acagacagga ccccggcctg 180
ggcctgcggc tgatctactt cagctacgac gtgaagatga aggaaaaggg cgacatcccc 240
gagggctaca gcgtgtccag agagaagaaa gagcggttca gcctgatcct ggaaagcgcc 300
agcaccaacc agaccagcat gtacctgtgc gccagcagcc ctttcggcag aggcagctac 360
gagcagtact tcggccctgg caccagactg accgtgaccg aagatctgaa gaacgtcttc 420
ccccctgagg tggcagtctt tgaaccctct gaggccgaaa tcagtcacac tcagaaagcc 480
accctggtgt gcctggctac agggttttac cctgatcacg tggagctgag ttggtgggtc 540
aacggcaagg aagtgcattc aggggtctgc actgacccac agcccctgaa agagcagcca 600
gcactgaatg attccaggta ctgtctgagt tcacggctga gagtgtctgc cacattttgg 660
cagaaccccc gaaatcattt ccggtgccag gtccagtttt atggcctgag cgagaatgac 720
gaatggacac aggatcgggc taagcccgtg actcagattg tctccgccga ggcttggggg 780
agagcagact gtggattcac ctccgaatct taccagcagg gcgtgctgtc tgccaccatc 840
ctgtacgaga ttctgctggg gaaggctaca ctgtatgccg tgctggtgag cgccctggtg 900
ctgatggcaa tggtcaagag aaaagacagc aggggaggct ctggcgccac caactttagc 960
ctgctgaagc aggccggcga tgtggaggag aatcctggcc caatgagcct gagcagcctg 1020
ctgaaggtgg tcaccgcttc tctgtggctg ggccctggaa tcgcccagaa gatcacccag 1080
acccagcccg gcatgttcgt gcaggaaaaa gaggccgtca ccctggactg cacctacgac 1140
accagcgacc agagctacgg cctgttctgg tacaagcagc ccagcagcgg cgagatgatc 1200
ttcctgatct accagggcag ctacgacgag cagaacgcca ccgagggccg gtacagcctg 1260
aacttccaga aggcccggaa gtccgccaac ctggtcatct ctgccagcca gctgggcgac 1320
agcgccatgt acttttgcgc catgagagag ggcagaggcg acagctgggg caagctgcag 1380
tttggagccg gcacccaggt ggtcgtgacc cctgatatcc agaatcccga ccctgccgtc 1440
taccagctga gggactcaaa gagctccgat aaaagcgtgt gcctgttcac cgactttgat 1500
tctcagacaa acgtcagcca gtccaaggac agtgacgtgt acattacaga caaatgcgtg 1560
ctggatatgc gctccatgga cttcaagtct aacagtgcag tggcctggtc caacaagtct 1620
gatttcgctt gcgcaaacgc ctttaacaat tcaatcattc cagaggatac cttctttcca 1680
agccccgaat ctagttgtga cgtgaagctg gtcgagaaat ctttcgaaac agatactaac 1740
ctgaattttc agaatctgag cgtgatcggc ttccgcattc tgctgctgaa agtggctggg 1800
tttaatctgc tgatgactct gcggctgtgg tcttcctaa 1839
<210> 442
<211> 1836
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 25 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 442
atgggcatca gactgctgtg cagagtggcc ttctgcttcc tggccgtggg cctggtggac 60
gtgaaagtga cccagagcag cagatacctg gtcaagcgga ccggcgagaa ggtgttcctg 120
gaatgcgtgc aggacatgga ccacgagaat atgttctggt acagacagga ccccggcctg 180
ggcctgcggc tgatctactt cagctacgac gtgaagatga aggaaaaggg cgacatcccc 240
gagggctaca gcgtgtccag agagaagaaa gagcggttca gcctgatcct ggaaagcgcc 300
agcaccaacc agaccagcat gtacctgtgc gccagcagcc tgtggggcca gagcaatcag 360
cctcagcact tcggcgacgg cacccggctg tctattctcg aggacctgaa taaggtgttc 420
ccccctgagg tggccgtgtt tgagccaagc gaggccgaga tctcccacac ccagaaggcc 480
accctggtgt gcctggcaac cggcttcttt cccgatcacg tggagctgtc ctggtgggtg 540
aacggcaagg aggtgcactc tggcgtgtgc acagacccac agcccctgaa ggagcagcct 600
gccctgaatg attcccgcta ttgtctgtcc tctcggctga gagtgtctgc caccttttgg 660
cagaacccac ggaatcactt cagatgccag gtgcagtttt acggcctgtc tgagaacgac 720
gagtggaccc aggatcgggc caagcctgtg acacagatcg tgagcgcgga agcatggggc 780
agagccgact gtggcttcac cagcgtgtcc tatcagcagg gcgtgctgtc cgccaccatc 840
ctgtacgaga tcctgctggg caaggccaca ctgtatgccg tgctggtgtc tgccctggtg 900
ctgatggcca tggtgaagag aaaagacttc ggctctggcg ccaccaactt tagcctgctg 960
aagcaggccg gcgatgtgga ggagaatcct ggcccaatga gcctgagcag cctgctgaag 1020
gtggtcaccg cttctctgtg gctgggccct ggaatcgccc agaagatcac ccagacccag 1080
cccggcatgt tcgtgcagga aaaagaggcc gtcaccctgg actgcaccta cgacaccagc 1140
gaccagagct acggcctgtt ctggtacaag cagcccagca gcggcgagat gatcttcctg 1200
atctaccagg gcagctacga cgagcagaac gccaccgagg gccggtacag cctgaacttc 1260
cagaaggccc ggaagtccgc caacctggtc atctctgcca gccagctggg cgacagcgcc 1320
atgtactttt gcgccatgag agagggcagc ctgaccggcg gaggcaacaa gctgaccttt 1380
ggcaccggca cccagctgaa agtggaactg aacatccaga atcccgaccc tgccgtctac 1440
cagctgaggg actcaaagag ctccgataaa agcgtgtgcc tgttcaccga ctttgattct 1500
cagacaaacg tcagccagtc caaggacagt gacgtgtaca ttacagacaa atgcgtgctg 1560
gatatgcgct ccatggactt caagtctaac agtgcagtgg cctggtccaa caagtctgat 1620
ttcgcttgcg caaacgcctt taacaattca atcattccag aggatacctt ctttccaagc 1680
cccgaatcta gttgtgacgt gaagctggtc gagaaatctt tcgaaacaga tactaacctg 1740
aattttcaga atctgagcgt gatcggcttc cgcattctgc tgctgaaagt ggctgggttt 1800
aatctgctga tgactctgcg gctgtggtct tcctaa 1836
<210> 443
<211> 1833
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 26 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 443
atgtctctgg gactgctgtg ctgcggagcc ttctctctgc tgtgggccgg acctgtgaat 60
gccggcgtga cccagacccc taagttccgg gtgctgaaaa ccggccagag catgaccctg 120
ctgtgcgccc aggacatgaa ccacgagtac atgtattggt acagacagga ccccggcatg 180
ggcctgcggc tgatccacta ttctgtgggc gagggcacca ccgccaaggg cgaagtgcct 240
gacggctaca acgtgtcccg gctgaagaag cagaacttcc tgctgggcct ggaaagcgcc 300
gctcctagcc agaccagcgt gtacttctgc gccagcagct actacgccag cggccggaac 360
tacgagcagt acttcggccc tggcacccgg ctgacagtga cagaagatct gaagaacgtc 420
ttcccccctg aggtggcagt ctttgaaccc tctgaggccg aaatcagtca cactcagaaa 480
gccaccctgg tgtgcctggc tacagggttt taccctgatc acgtggagct gagttggtgg 540
gtcaacggca aggaagtgca ttcaggggtc tgcactgacc cacagcccct gaaagagcag 600
ccagcactga atgattccag gtactgtctg agttcacggc tgagagtgtc tgccacattt 660
tggcagaacc cccgaaatca tttccggtgc caggtccagt tttatggcct gagcgagaat 720
gacgaatgga cacaggatcg ggctaagccc gtgactcaga ttgtctccgc cgaggcttgg 780
gggagagcag actgtggatt cacctccgaa tcttaccagc agggcgtgct gtctgccacc 840
atcctgtacg agattctgct ggggaaggct acactgtatg ccgtgctggt gagcgccctg 900
gtgctgatgg caatggtcaa gagaaaagac agcaggggag gctctggcgc caccaacttt 960
agcctgctga agcaggccgg cgatgtggag gagaatcctg gcccaatgag cctgagcagc 1020
ctgctgaagg tggtcaccgc ttctctgtgg ctgggccctg gaatcgccca gaagatcacc 1080
cagacccagc ccggcatgtt cgtgcaggaa aaagaggccg tcaccctgga ctgcacctac 1140
gacaccagcg accagagcta cggcctgttc tggtacaagc agcccagcag cggcgagatg 1200
atcttcctga tctaccaggg cagctacgac gagcagaacg ccaccgaggg ccggtacagc 1260
ctgaacttcc agaaggcccg gaagtccgcc aacctggtca tctctgccag ccagctgggc 1320
gacagcgcca tgtacttttg cgccatgcgg gacgcccgga acaacgacat gagattcgga 1380
gccggcaccc ggctgaccgt gaagcctaat atccagaatc ccgaccctgc cgtctaccag 1440
ctgagggact caaagagctc cgataaaagc gtgtgcctgt tcaccgactt tgattctcag 1500
acaaacgtca gccagtccaa ggacagtgac gtgtacatta cagacaaatg cgtgctggat 1560
atgcgctcca tggacttcaa gtctaacagt gcagtggcct ggtccaacaa gtctgatttc 1620
gcttgcgcaa acgcctttaa caattcaatc attccagagg ataccttctt tccaagcccc 1680
gaatctagtt gtgacgtgaa gctggtcgag aaatctttcg aaacagatac taacctgaat 1740
tttcagaatc tgagcgtgat cggcttccgc attctgctgc tgaaagtggc tgggtttaat 1800
ctgctgatga ctctgcggct gtggtcttcc taa 1833
<210> 444
<211> 1857
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 27 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 444
atgcacagac ctagaaggcc cctgcatccc gtggcccctg ccatgtctat cggcctgctg 60
tgctgcgtgg cattctccct gctgtgggcc tctcctgtga atgccggcgt gacccagacc 120
cctaagttcc aggtgctgaa aaccggccag agcatgaccc tgcagtgcgc ccaggacatg 180
aaccacaaca gcatgtactg gtacagacag gaccccggca tgggcctgcg gctgatctac 240
tatagcgcca gcgagggcac caccgacaag ggcgaggtgc ccaacggcta caacgtgtcc 300
cggctgaaca agagagagtt cagcctgaga ctggaaagcg ccgctcccag ccagaccagc 360
gtgtactttt gcgccagcag cgagttcggc agcctgaacg agaagctgtt cttcggcagc 420
ggcacccagc tgagcgtgct cgaggacctg aataaggtgt tcccccctga ggtggccgtg 480
tttgagccaa gcgaggccga gatctcccac acccagaagg ccaccctggt gtgcctggca 540
accggcttct ttcccgatca cgtggagctg tcctggtggg tgaacggcaa ggaggtgcac 600
tctggcgtgt gcacagaccc acagcccctg aaggagcagc ctgccctgaa tgattcccgc 660
tattgtctgt cctctcggct gagagtgtct gccacctttt ggcagaaccc acggaatcac 720
ttcagatgcc aggtgcagtt ttacggcctg tctgagaacg acgagtggac ccaggatcgg 780
gccaagcctg tgacacagat cgtgagcgcg gaagcatggg gcagagccga ctgtggcttc 840
accagcgtgt cctatcagca gggcgtgctg tccgccacca tcctgtacga gatcctgctg 900
ggcaaggcca cactgtatgc cgtgctggtg tctgccctgg tgctgatggc catggtgaag 960
agaaaagact tcggctctgg cgccaccaac tttagcctgc tgaagcaggc cggcgatgtg 1020
gaggagaatc ctggcccaat gaagctggtc accagcatca ccgtgctgct gagcctggga 1080
atcatgggcg acgccaagac cacccagccc aacagcatgg aaagcaacga agaggaaccc 1140
gtgcatctgc cctgcaacca cagcaccatc agcggcaccg actacatcca ctggtacaga 1200
cagctgccca gccagggccc cgagtatgtg atccacggcc tgaccagcaa cgtgaacaac 1260
cggatggcct ccctggccat tgccgaggac agaaagagca gcaccctgat cctgcaccgg 1320
gccacactga gagatgccgc cgtgtactac tgcatcctgc gggtgccacc tcagagcggc 1380
ggctaccaga aagtgacctt cggcaccggc accaagctgc aggtcatccc caatatccag 1440
aatcccgacc ctgccgtcta ccagctgagg gactcaaaga gctccgataa aagcgtgtgc 1500
ctgttcaccg actttgattc tcagacaaac gtcagccagt ccaaggacag tgacgtgtac 1560
attacagaca aatgcgtgct ggatatgcgc tccatggact tcaagtctaa cagtgcagtg 1620
gcctggtcca acaagtctga tttcgcttgc gcaaacgcct ttaacaattc aatcattcca 1680
gaggatacct tctttccaag ccccgaatct agttgtgacg tgaagctggt cgagaaatct 1740
ttcgaaacag atactaacct gaattttcag aatctgagcg tgatcggctt ccgcattctg 1800
ctgctgaaag tggctgggtt taatctgctg atgactctgc ggctgtggtc ttcctaa 1857
<210> 445
<211> 1827
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 28 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 445
atgggcatca gactgctgtg cagagtggcc ttctgcttcc tggccgtggg cctggtggac 60
gtgaaagtga cccagagcag cagatacctg gtcaagcgga ccggcgagaa ggtgttcctg 120
gaatgcgtgc aggacatgga ccacgagaat atgttctggt acagacagga ccccggcctg 180
ggcctgcggc tgatctactt cagctacgac gtgaagatga aggaaaaggg cgacatcccc 240
gagggctaca gcgtgtccag agagaagaaa gagcggttca gcctgatcct ggaaagcgcc 300
agcaccaacc agaccagcat gtacctgtgc gccagcagcc tgtggggcag aagcagcggc 360
aacaccatct acttcggcga gggcagctgg ctgaccgtgg tcgaggacct gaataaggtg 420
ttcccccctg aggtggccgt gtttgagcca agcgaggccg agatctccca cacccagaag 480
gccaccctgg tgtgcctggc aaccggcttc tttcccgatc acgtggagct gtcctggtgg 540
gtgaacggca aggaggtgca ctctggcgtg tgcacagacc cacagcccct gaaggagcag 600
cctgccctga atgattcccg ctattgtctg tcctctcggc tgagagtgtc tgccaccttt 660
tggcagaacc cacggaatca cttcagatgc caggtgcagt tttacggcct gtctgagaac 720
gacgagtgga cccaggatcg ggccaagcct gtgacacaga tcgtgagcgc ggaagcatgg 780
ggcagagccg actgtggctt caccagcgtg tcctatcagc agggcgtgct gtccgccacc 840
atcctgtacg agatcctgct gggcaaggcc acactgtatg ccgtgctggt gtctgccctg 900
gtgctgatgg ccatggtgaa gagaaaagac ttcggctctg gcgccaccaa ctttagcctg 960
ctgaagcagg ccggcgatgt ggaggagaat cctggcccaa tgatgatcag cctgcgggtg 1020
ctgctggtca tcctgtggct gcagctgagc tgggtctggt cccagcggaa agaggtggaa 1080
caggaccctg gccccttcaa cgtgccagag ggcgctaccg tggccttcaa ctgcacctac 1140
agcaacagcg ccagccagag cttcttctgg tacagacagg actgccggaa agaacccaag 1200
ctgctgatga gcgtgtacag cagcggcaac gaggacggca gattcaccgc ccagctgaac 1260
cgggccagcc agtacatctc cctgctgatc cgggacagca agctgagcga cagcgccacc 1320
tatctgtgtg tcgtgcgggg aggcggcacc agctacggca agctgacatt tggccagggc 1380
accatcctga ccgtgcaccc caatatccag aatcccgacc ctgccgtcta ccagctgagg 1440
gactcaaaga gctccgataa aagcgtgtgc ctgttcaccg actttgattc tcagacaaac 1500
gtcagccagt ccaaggacag tgacgtgtac attacagaca aatgcgtgct ggatatgcgc 1560
tccatggact tcaagtctaa cagtgcagtg gcctggtcca acaagtctga tttcgcttgc 1620
gcaaacgcct ttaacaattc aatcattcca gaggatacct tctttccaag ccccgaatct 1680
agttgtgacg tgaagctggt cgagaaatct ttcgaaacag atactaacct gaattttcag 1740
aatctgagcg tgatcggctt ccgcattctg ctgctgaaag tggctgggtt taatctgctg 1800
atgactctgc ggctgtggtc ttcctaa 1827
<210> 446
<211> 1836
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 29 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 446
atgagcaatc aggtgctgtg ctgcgtggtc ctgtgcctgc tgggagccaa taccgtggat 60
ggcggcatca cccagagccc caagtacctg ttccggaaag agggccagaa cgtcaccctg 120
agctgcgagc agaacctgaa ccacgacgcc atgtactggt acagacagga ccccggccag 180
ggcctgcggc tgatctacta cagccagatc gtgaacgact tccagaaggg cgacattgcc 240
gagggctaca gcgtgtccag agagaagaaa gagtccttcc cactgaccgt gaccagcgcc 300
cagaagaacc ccaccgcctt ctacctgtgt gccagctctc cttggggcag agccaccaac 360
gagcagttct tcggccctgg caccaggctg acagtgctgg aagatctgaa gaacgtcttc 420
ccccctgagg tggcagtctt tgaaccctct gaggccgaaa tcagtcacac tcagaaagcc 480
accctggtgt gcctggctac agggttttac cctgatcacg tggagctgag ttggtgggtc 540
aacggcaagg aagtgcattc aggggtctgc actgacccac agcccctgaa agagcagcca 600
gcactgaatg attccaggta ctgtctgagt tcacggctga gagtgtctgc cacattttgg 660
cagaaccccc gaaatcattt ccggtgccag gtccagtttt atggcctgag cgagaatgac 720
gaatggacac aggatcgggc taagcccgtg actcagattg tctccgccga ggcttggggg 780
agagcagact gtggattcac ctccgaatct taccagcagg gcgtgctgtc tgccaccatc 840
ctgtacgaga ttctgctggg gaaggctaca ctgtatgccg tgctggtgag cgccctggtg 900
ctgatggcaa tggtcaagag aaaagacagc aggggaggct ctggcgccac caactttagc 960
ctgctgaagc aggccggcga tgtggaggag aatcctggcc caatgagcct gagcagcctg 1020
ctgaaggtgg tcaccgcttc tctgtggctg ggccctggaa tcgcccagaa gatcacccag 1080
acccagcccg gcatgttcgt gcaggaaaaa gaggccgtca ccctggactg cacctacgac 1140
accagcgacc agagctacgg cctgttctgg tacaagcagc ccagcagcgg cgagatgatc 1200
ttcctgatct accagggcag ctacgacgag cagaacgcca ccgagggccg gtacagcctg 1260
aacttccaga aggcccggaa gtccgccaac ctggtcatct ctgccagcca gctgggcgac 1320
agcgccatgt acttttgcgc catgcggctg aacaccggca ccgccagcaa gctgaccttc 1380
ggcacaggca ccagactgca ggtcacactg gacatccaga atcccgaccc tgccgtctac 1440
cagctgaggg actcaaagag ctccgataaa agcgtgtgcc tgttcaccga ctttgattct 1500
cagacaaacg tcagccagtc caaggacagt gacgtgtaca ttacagacaa atgcgtgctg 1560
gatatgcgct ccatggactt caagtctaac agtgcagtgg cctggtccaa caagtctgat 1620
ttcgcttgcg caaacgcctt taacaattca atcattccag aggatacctt ctttccaagc 1680
cccgaatcta gttgtgacgt gaagctggtc gagaaatctt tcgaaacaga tactaacctg 1740
aattttcaga atctgagcgt gatcggcttc cgcattctgc tgctgaaagt ggctgggttt 1800
aatctgctga tgactctgcg gctgtggtct tcctaa 1836
<210> 447
<211> 1818
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 30 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 447
atggatacct ggctcgtgtg ctgggccatc ttcagcctgc tgaaggccgg actgaccgag 60
cccgaagtga cccagacacc cagccaccag gtcacacaga tgggccagga agtgatcctg 120
cgctgcgtgc ccatcagcaa ccacctgtac ttctactggt acagacagat cctgggccag 180
aaggtggaat tcctcgtcag cttctacaac aacgagatca gcgagaagtc cgagattttc 240
gacgaccagt tcagcgtgga acggcccgac ggcagcaact tcaccctgaa gatcagaagc 300
accaagctcg aggacagcgc catgtacttt tgcgccagca gcagacagcc cagcagcggc 360
aacaccatct acttcggcga gggcagctgg ctgaccgtgg tcgaggacct gaataaggtg 420
ttcccccctg aggtggccgt gtttgagcca agcgaggccg agatctccca cacccagaag 480
gccaccctgg tgtgcctggc aaccggcttc tttcccgatc acgtggagct gtcctggtgg 540
gtgaacggca aggaggtgca ctctggcgtg tgcacagacc cacagcccct gaaggagcag 600
cctgccctga atgattcccg ctattgtctg tcctctcggc tgagagtgtc tgccaccttt 660
tggcagaacc cacggaatca cttcagatgc caggtgcagt tttacggcct gtctgagaac 720
gacgagtgga cccaggatcg ggccaagcct gtgacacaga tcgtgagcgc ggaagcatgg 780
ggcagagccg actgtggctt caccagcgtg tcctatcagc agggcgtgct gtccgccacc 840
atcctgtacg agatcctgct gggcaaggcc acactgtatg ccgtgctggt gtctgccctg 900
gtgctgatgg ccatggtgaa gagaaaagac ttcggctctg gcgccaccaa ctttagcctg 960
ctgaagcagg ccggcgatgt ggaggagaat cctggcccaa tgcggctggt ggccagagtg 1020
accgtgttcc tgaccttcgg caccatcatc gacgccaaga ccacccagcc tcccagcatg 1080
gattgtgccg agggcagagc cgccaacctg ccctgcaatc acagcaccat cagcggcaac 1140
gagtacgtgt actggtacag acagatccac agccagggcc ctcagtacat catccacggc 1200
ctgaagaaca acgagacaaa cgagatggcc agcctgatca tcaccgagga cagaaagagc 1260
agcaccctga tcctgcccca cgccaccctg agagacaccg ccgtgtacta ctgtatcgtg 1320
cggggcacaa gcgtgctgca gggcaacgag aagctgacat ttggcaccgg cacccggctg 1380
accatcatcc ccaatatcca gaatcccgac cctgccgtct accagctgag ggactcaaag 1440
agctccgata aaagcgtgtg cctgttcacc gactttgatt ctcagacaaa cgtcagccag 1500
tccaaggaca gtgacgtgta cattacagac aaatgcgtgc tggatatgcg ctccatggac 1560
ttcaagtcta acagtgcagt ggcctggtcc aacaagtctg atttcgcttg cgcaaacgcc 1620
tttaacaatt caatcattcc agaggatacc ttctttccaa gccccgaatc tagttgtgac 1680
gtgaagctgg tcgagaaatc tttcgaaaca gatactaacc tgaattttca gaatctgagc 1740
gtgatcggct tccgcattct gctgctgaaa gtggctgggt ttaatctgct gatgactctg 1800
cggctgtggt cttcctaa 1818
<210> 448
<211> 1845
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 31 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 448
atgggcacca gactgctgtt ctgggtggcc ttctgcctgc tgggagccga tcatacaggt 60
gccggcgtgt cccagagccc cagcaacaaa gtgaccgaga agggcaagga cgtggaactg 120
agatgcgacc ccatcagcgg ccacaccgcc ctgtactggt acagacagtc tctgggccag 180
ggcctggaat tcctgatcta ttttcagggc aacagcgccc ctgacaagag cggcctgccc 240
agcgatagat tcagcgccga gagaacaggc ggcagcgtgt ccaccctgac catccagaga 300
acccagcagg aagatagcgc cgtctacctg tgcgccagca gcagattcct gggcagcacc 360
gacacccagt acttcggccc aggcaccagg ctgacagtgc tggaagatct gaagaacgtc 420
ttcccccctg aggtggcagt ctttgaaccc tctgaggccg aaatcagtca cactcagaaa 480
gccaccctgg tgtgcctggc tacagggttt taccctgatc acgtggagct gagttggtgg 540
gtcaacggca aggaagtgca ttcaggggtc tgcactgacc cacagcccct gaaagagcag 600
ccagcactga atgattccag gtactgtctg agttcacggc tgagagtgtc tgccacattt 660
tggcagaacc cccgaaatca tttccggtgc caggtccagt tttatggcct gagcgagaat 720
gacgaatgga cacaggatcg ggctaagccc gtgactcaga ttgtctccgc cgaggcttgg 780
gggagagcag actgtggatt cacctccgaa tcttaccagc agggcgtgct gtctgccacc 840
atcctgtacg agattctgct ggggaaggct acactgtatg ccgtgctggt gagcgccctg 900
gtgctgatgg caatggtcaa gagaaaagac agcaggggag gctctggcgc caccaacttt 960
agcctgctga agcaggccgg cgatgtggag gagaatcctg gcccaatggc catgctgctg 1020
ggagcctctg tgctgatcct gtggctccag cccgactggg tcaacagcca gcagaagaac 1080
gacgaccagc aggtcaaaca gaacagcccc agcctgagcg tgcaggaagg ccggatcagc 1140
atcctgaact gcgactacac caacagcatg ttcgactact ttctgtggta caagaagtac 1200
cccgccgagg gccccacctt cctgatctcc atcagcagca tcaaggacaa gaacgaggac 1260
ggccggttca ccgtgtttct gaacaagagc gccaagcacc tgagcctgca catcgtgcct 1320
agccagcctg gcgacagcgc cgtgtacttt tgtgccgcca gcgagcgggg cacctacaag 1380
tacatcttcg gcaccggcac ccggctgaag gtgctggcta atatccagaa tcccgaccct 1440
gccgtctacc agctgaggga ctcaaagagc tccgataaaa gcgtgtgcct gttcaccgac 1500
tttgattctc agacaaacgt cagccagtcc aaggacagtg acgtgtacat tacagacaaa 1560
tgcgtgctgg atatgcgctc catggacttc aagtctaaca gtgcagtggc ctggtccaac 1620
aagtctgatt tcgcttgcgc aaacgccttt aacaattcaa tcattccaga ggataccttc 1680
tttccaagcc ccgaatctag ttgtgacgtg aagctggtcg agaaatcttt cgaaacagat 1740
actaacctga attttcagaa tctgagcgtg atcggcttcc gcattctgct gctgaaagtg 1800
gctgggttta atctgctgat gactctgcgg ctgtggtctt cctaa 1845
<210> 449
<211> 1809
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 32 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 449
atgggcttca gactgctgtg ctgcgtggcc ttctgtctgc tgggagccgg acctgtggat 60
agcggcgtga cccagacccc taagcacctg atcaccgcca ccggccagcg cgtgacactg 120
agatgtagcc ctagaagcgg cgacctgagc gtgtactggt atcagcagag cctggaccag 180
ggcctgcagt tcctgatcca gtactacaac ggcgaggaac gggccaaggg caacatcctg 240
gaacggttca gcgcccagca gttccccgat ctgcacagcg agctgaacct gagcagcctg 300
gaactgggcg acagcgccct gtacttctgt gcctcttctg tgggcggcga ccacagcgac 360
gagcagtttt ttggccctgg cacccggctg accgtgctgg aagatctgaa gaacgtcttc 420
ccccctgagg tggcagtctt tgaaccctct gaggccgaaa tcagtcacac tcagaaagcc 480
accctggtgt gcctggctac agggttttac cctgatcacg tggagctgag ttggtgggtc 540
aacggcaagg aagtgcattc aggggtctgc actgacccac agcccctgaa agagcagcca 600
gcactgaatg attccaggta ctgtctgagt tcacggctga gagtgtctgc cacattttgg 660
cagaaccccc gaaatcattt ccggtgccag gtccagtttt atggcctgag cgagaatgac 720
gaatggacac aggatcgggc taagcccgtg actcagattg tctccgccga ggcttggggg 780
agagcagact gtggattcac ctccgaatct taccagcagg gcgtgctgtc tgccaccatc 840
ctgtacgaga ttctgctggg gaaggctaca ctgtatgccg tgctggtgag cgccctggtg 900
ctgatggcaa tggtcaagag aaaagacagc aggggaggct ctggcgccac caactttagc 960
ctgctgaagc aggccggcga tgtggaggag aatcctggcc caatggtgct gaagttcagc 1020
gtgtccatcc tgtggatcca gctggcctgg gtgtccaccc agctgctgga acagagccct 1080
cagttcctga gcatccagga aggcgagaac ctgaccgtgt actgcaacag cagcagcgtg 1140
ttcagcagcc tgcagtggta cagacaggaa cccggcgagg gacctgtgct gctggtcaca 1200
gtcgtgacag gcggcgaagt gaagaagctg aagcggctga ccttccagtt cggcgacgcc 1260
agaaaggaca gctccctgca cattacagcc gcccagcctg gcgataccgg cctgtatctg 1320
tgtgccggcg gaagcaacta caagctgacc tttggcaagg gcaccctgct gaccgtgaac 1380
cccaatatcc agaatcccga ccctgccgtc taccagctga gggactcaaa gagctccgat 1440
aaaagcgtgt gcctgttcac cgactttgat tctcagacaa acgtcagcca gtccaaggac 1500
agtgacgtgt acattacaga caaatgcgtg ctggatatgc gctccatgga cttcaagtct 1560
aacagtgcag tggcctggtc caacaagtct gatttcgctt gcgcaaacgc ctttaacaat 1620
tcaatcattc cagaggatac cttctttcca agccccgaat ctagttgtga cgtgaagctg 1680
gtcgagaaat ctttcgaaac agatactaac ctgaattttc agaatctgag cgtgatcggc 1740
ttccgcattc tgctgctgaa agtggctggg tttaatctgc tgatgactct gcggctgtgg 1800
tcttcctaa 1809
<210> 450
<211> 1830
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 33 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 450
atggatacct ggctcgtgtg ctgggccatc ttcagcctgc tgaaggccgg actgaccgag 60
cccgaagtga cccagacacc cagccaccag gtcacacaga tgggccagga agtgatcctg 120
cgctgcgtgc ccatcagcaa ccacctgtac ttctactggt acagacagat cctgggccag 180
aaggtggaat tcctcgtcag cttctacaac aacgagatca gcgagaagtc cgagattttc 240
gacgaccagt tcagcgtgga acggcccgac ggcagcaact tcaccctgaa gatcagaagc 300
accaagctcg aggacagcgc catgtacttt tgcgccagca cccctcggga cacctacgag 360
cagtacttcg gccctggcac cagactgacc gtgaccgaag atctgaagaa cgtcttcccc 420
cctgaggtgg cagtctttga accctctgag gccgaaatca gtcacactca gaaagccacc 480
ctggtgtgcc tggctacagg gttttaccct gatcacgtgg agctgagttg gtgggtcaac 540
ggcaaggaag tgcattcagg ggtctgcact gacccacagc ccctgaaaga gcagccagca 600
ctgaatgatt ccaggtactg tctgagttca cggctgagag tgtctgccac attttggcag 660
aacccccgaa atcatttccg gtgccaggtc cagttttatg gcctgagcga gaatgacgaa 720
tggacacagg atcgggctaa gcccgtgact cagattgtct ccgccgaggc ttgggggaga 780
gcagactgtg gattcacctc cgaatcttac cagcagggcg tgctgtctgc caccatcctg 840
tacgagattc tgctggggaa ggctacactg tatgccgtgc tggtgagcgc cctggtgctg 900
atggcaatgg tcaagagaaa agacagcagg ggaggctctg gcgccaccaa ctttagcctg 960
ctgaagcagg ccggcgatgt ggaggagaat cctggcccaa tgatgaagtc cctgcgggtg 1020
ctgctggtca tcctgtggct gcagctgagc tgggtctggt cccagcagaa agaggtggaa 1080
cagaacagcg gcccactgag cgtgccagag ggcgctatcg ccagcctgaa ctgcacctac 1140
agcgaccggg gcagccagag cttcttctgg tacagacagt acagcggcaa gagccccgag 1200
ctgatcatgt tcatctacag caacggcgac aaagaggacg gccggttcac cgcccagctg 1260
aacaaggcca gccagtacgt gtccctgctg atcagagaca gccagcccag cgacagcgcc 1320
acctacctgt gtgccgtgaa tgcccaccac accggcggct tcaagaccat ctttggagcc 1380
ggcacccggc tgttcgtgaa ggccaatatc cagaatcccg accctgccgt ctaccagctg 1440
agggactcaa agagctccga taaaagcgtg tgcctgttca ccgactttga ttctcagaca 1500
aacgtcagcc agtccaagga cagtgacgtg tacattacag acaaatgcgt gctggatatg 1560
cgctccatgg acttcaagtc taacagtgca gtggcctggt ccaacaagtc tgatttcgct 1620
tgcgcaaacg cctttaacaa ttcaatcatt ccagaggata ccttctttcc aagccccgaa 1680
tctagttgtg acgtgaagct ggtcgagaaa tctttcgaaa cagatactaa cctgaatttt 1740
cagaatctga gcgtgatcgg cttccgcatt ctgctgctga aagtggctgg gtttaatctg 1800
ctgatgactc tgcggctgtg gtcttcctaa 1830
<210> 451
<211> 1806
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 34 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 451
atgggacctg gactgctgtg ttgggccctg ctgtgtctgc tgggagccgg atctgtggaa 60
accggcgtga cccagagccc cacccacctg atcaagacca gaggccagca ggtcacactg 120
cggtgtagca gccagagcgg ccacaacacc gtgtcctggt atcagcaggc cctgggccag 180
ggaccccagt tcatcttcca gtactaccgc gaggaagaga acggcagagg caacttccca 240
ccccggttca gcggcctgca gttccccaat tacagcagcg agctgaacgt gaacgccctg 300
gaactggacg acagcgccct gtacctgtgc gcctctagca gctacgccgg cagctacgag 360
cagtacttcg gccctggcac cagactgacc gtgaccgaag atctgaagaa cgtcttcccc 420
cctgaggtgg cagtctttga accctctgag gccgaaatca gtcacactca gaaagccacc 480
ctggtgtgcc tggctacagg gttttaccct gatcacgtgg agctgagttg gtgggtcaac 540
ggcaaggaag tgcattcagg ggtctgcact gacccacagc ccctgaaaga gcagccagca 600
ctgaatgatt ccaggtactg tctgagttca cggctgagag tgtctgccac attttggcag 660
aacccccgaa atcatttccg gtgccaggtc cagttttatg gcctgagcga gaatgacgaa 720
tggacacagg atcgggctaa gcccgtgact cagattgtct ccgccgaggc ttgggggaga 780
gcagactgtg gattcacctc cgaatcttac cagcagggcg tgctgtctgc caccatcctg 840
tacgagattc tgctggggaa ggctacactg tatgccgtgc tggtgagcgc cctggtgctg 900
atggcaatgg tcaagagaaa agacagcagg ggaggctctg gcgccaccaa ctttagcctg 960
ctgaagcagg ccggcgatgt ggaggagaat cctggcccaa tgaagaaact gctggccatg 1020
atcctgtggc tgcagctgga cagactgagc ggcgagctga aggtggaaca gaaccctctg 1080
ttcctgagca tgcaggaagg caagaactac accatctact gcaactacag caccaccagc 1140
gaccggctgt actggtacag acaggacccc ggcaagagcc tggaaagcct gttcgtgctg 1200
ctgagcaacg gggccgtgaa gcaggaagga cggctgatgg ccagcctgga caccaaggcc 1260
agactgtcca ccctgcacat cacagccgcc gtgcacgacc tgagcgccac ctacttttgt 1320
gccgtgtccg gcacctacaa gtacatcttc ggcaccggca ccagactgaa ggtgctggcc 1380
aatatccaga atcccgaccc tgccgtctac cagctgaggg actcaaagag ctccgataaa 1440
agcgtgtgcc tgttcaccga ctttgattct cagacaaacg tcagccagtc caaggacagt 1500
gacgtgtaca ttacagacaa atgcgtgctg gatatgcgct ccatggactt caagtctaac 1560
agtgcagtgg cctggtccaa caagtctgat ttcgcttgcg caaacgcctt taacaattca 1620
atcattccag aggatacctt ctttccaagc cccgaatcta gttgtgacgt gaagctggtc 1680
gagaaatctt tcgaaacaga tactaacctg aattttcaga atctgagcgt gatcggcttc 1740
cgcattctgc tgctgaaagt ggctgggttt aatctgctga tgactctgcg gctgtggtct 1800
tcctaa 1806
<210> 452
<211> 1818
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 35 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 452
atgggcttta gactgctgtg ctgcgtggcc ttctgtctgc ttggagctgg ccctgtggat 60
agcggcgtta cccagacacc taagcacctg atcacagcca caggccagcg cgtgaccctg 120
agatgttctc ctagaagcgg cgacctgagc gtgtactggt atcagcagtc tctggaccag 180
ggcctgcagt tcctgatcca gtactacaac ggcgaggaaa gagccaaggg caacatcctg 240
gaacggttca gcgcccagca gttcccagat ctgcacagcg agctgaacct gagcagcctg 300
gaactgggag atagcgccct gtacttctgt gccagcacca caagcggcga tagcagctac 360
aacgagcagt tcttcggccc tggcaccaga ctgacagttc tggaagatct gaagaacgtc 420
ttcccacctg aggtggccgt gttcgagcct tctgaggccg agatcagcca cacacagaaa 480
gccacactcg tgtgtctggc caccggcttc tatcccgatc acgtggaact gtcttggtgg 540
gtcaacggca aagaggtgca cagcggcgtc tgtaccgatc ctcagcctct gaaagagcag 600
cccgctctga acgacagcag atactgcctg agcagcagac tgagagtgtc cgccaccttc 660
tggcagaacc ccagaaacca cttcagatgc caggtgcagt tctacggcct gagcgagaac 720
gatgagtgga cccaggatag agccaagcct gtgacacaga tcgtgtctgc cgaagcctgg 780
ggcagagccg attgtggctt taccagcgag agctaccagc agggcgtgct gtctgccaca 840
atcctgtacg agatcctgct gggcaaagcc actctgtacg ccgtgctggt gtctgccctg 900
gtgctgatgg ccatggtcaa gcggaaggat agcagaggcg gaagcggcgc cacaaacttc 960
tcactgctga aacaggctgg cgacgtggag gagaatcctg gcccaatggc tctgcagtct 1020
acactgggag ctgtgtggct gggcctgctg ctgaattctc tgtggaaggt ggccgagagc 1080
aaggaccagg tgttccagcc ttctacagtg gcctcttctg agggcgccgt ggtggaaatc 1140
ttctgcaacc acagcgtgtc caacgcctac aacttcttct ggtatctgca cttccccggc 1200
tgcgccccta gactgcttgt gaagggaagc aagcctagcc agcagggcag atacaacatg 1260
acctacgagc ggttcagcag ctccctgctg attctgcaag tgcgggaagc cgatgccgcc 1320
gtgtattatt gtgctgtggc cggcgactac aagctgagct ttggagccgg caccacagtg 1380
acagtgcggg ctaatatcca gaatccggac cctgccgtct accagctgag ggactcaaag 1440
agctccgata aaagcgtgtg cctgttcacc gactttgatt ctcagacaaa cgtcagccag 1500
tccaaggaca gtgacgtgta cattacagac aaatgcgtgc tggatatgcg ctccatggac 1560
ttcaagtcta acagtgcagt ggcctggtcc aacaagtctg atttcgcttg cgcaaacgcc 1620
tttaacaatt caatcattcc agaggatacc ttctttccaa gccccgaatc tagttgtgac 1680
gtgaagctgg tcgagaaatc tttcgaaaca gatactaacc tgaattttca gaatctgagc 1740
gtgatcggct tccgcattct gctgctgaaa gtggctgggt ttaatctgct gatgactctg 1800
cggctgtggt cttcctaa 1818
<210> 453
<211> 1824
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 36 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 453
atggatactt ggcttgtgtg ctgggccatc ttcagcctgc tgaaggccgg actgacagag 60
cccgaagtga cacagacacc cagccaccaa gtgacccaga tgggccaaga agtgatcctg 120
cgctgcgtgc ccatcagcaa ccacctgtac ttctactggt acagacagat cctgggccag 180
aaagtggaat tcctggtgtc cttctacaac aacgagatca gcgagaagtc cgagatcttc 240
gacgaccagt tcagcgtgga aagacccgac ggcagcaact tcaccctgaa gatcagaagc 300
accaagctcg aggacagcgc catgtacttt tgcgccatga ccggcagaag caactacgag 360
cagtacttcg gccctggcac cagactgaca gtgacagaag atctgaagaa cgtcttccca 420
cctgaggtgg ccgtgttcga gccttctgag gccgagatca gccacacaca gaaagccaca 480
ctcgtgtgtc tggccaccgg cttctatccc gatcacgtgg aactgtcttg gtgggtcaac 540
ggcaaagagg tgcacagcgg cgtctgtacc gatcctcagc ctctgaaaga gcagcccgct 600
ctgaacgaca gcagatactg cctgagcagc agactgagag tgtccgccac cttctggcag 660
aaccccagaa accacttcag atgccaggtg cagttctacg gcctgagcga gaacgatgag 720
tggacccagg atagagccaa gcctgtgaca cagatcgtgt ctgccgaagc ctggggcaga 780
gccgattgtg gctttaccag cgagagctac cagcagggcg tgctgtctgc cacaatcctg 840
tacgagatcc tgctgggcaa agccactctg tacgccgtgc tggtgtctgc cctggtgctg 900
atggccatgg tcaagcggaa ggatagcaga ggcggaagcg gcgccacaaa cttctcactg 960
ctgaaacagg ctggcgacgt ggaggagaat cctggcccaa tgatgatcag cctgcgggtg 1020
ctgctggtca tcctgtggct gcaactgagc tgggtctggt cccagcggaa agaggtggaa 1080
caggaccccg gacctttcaa tgtgcctgaa ggcgccaccg tggccttcaa ctgcacctac 1140
agcaatagcg ccagccagag cttcttctgg tacagacagg actgccggaa agaacccaag 1200
ctgctgatga gcgtgtacag cagcggcaac gaggacggca gattcacagc ccagctgaac 1260
agagccagcc agtacatctc cctgctgatc cgggatagca agctgagcga tagcgccacc 1320
tacctgtgcg tggtcaacag agacaactac ggccagaact tcgtgttcgg ccctggcaca 1380
agactgagcg tgctgcctta tatccagaat ccggaccctg ccgtctacca gctgagggac 1440
tcaaagagct ccgataaaag cgtgtgcctg ttcaccgact ttgattctca gacaaacgtc 1500
agccagtcca aggacagtga cgtgtacatt acagacaaat gcgtgctgga tatgcgctcc 1560
atggacttca agtctaacag tgcagtggcc tggtccaaca agtctgattt cgcttgcgca 1620
aacgccttta acaattcaat cattccagag gataccttct ttccaagccc cgaatctagt 1680
tgtgacgtga agctggtcga gaaatctttc gaaacagata ctaacctgaa ttttcagaat 1740
ctgagcgtga tcggcttccg cattctgctg ctgaaagtgg ctgggtttaa tctgctgatg 1800
actctgcggc tgtggtcttc ctaa 1824
<210> 454
<211> 1812
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 37 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 454
atgggaacaa gactgctgtg ttgggtcgtg ctgggcttcc tgggcacaga tcatacaggt 60
gccggtgtct ctcagagccc cagatacaag gtggccaagc gcggacagga tgtggccctg 120
agatgtgatc ctatcagcgg ccacgtgtcc ctgttctggt atcagcaggc tctcggacag 180
ggccccgagt tcctgaccta ctttcagaat gaggcccagc tggacaagag cggcctgcct 240
agcgatagat tcttcgccga aagacccgag ggcagcgtgt ccacactgaa gatccagaga 300
acccagcaag aggacagcgc cgtgtacctg tgtgcctcta gtctgctgct gggcgcctac 360
ggctacacat ttggctctgg caccagactg accgtggtcg aggacctgaa taaggtgttc 420
cctccagagg tggccgtgtt cgagccttct gaggccgaga tcagccacac acagaaagcc 480
acactcgtgt gcctggccac cggctttttt cccgatcacg tggaactgtc ttggtgggtc 540
aacggcaaag aggtgcacag cggcgtctgt accgatcctc agcctctgaa agagcagccc 600
gctctgaacg acagcagata ctgcctgagc agcagactga gagtgtccgc caccttctgg 660
cagaacccca gaaaccactt cagatgccag gtgcagttct acggcctgag cgagaacgat 720
gagtggaccc aggatagagc caagcctgtg acacagatcg tgtctgccga agcctggggc 780
agagccgatt gtggctttac cagcgtgtcc tatcagcagg gcgtgctgtc tgccaccatc 840
ctgtatgaga tcctgctggg caaagccact ctgtacgccg tgctggtgtc tgccctggtg 900
ctgatggcca tggtcaagag aaaggacttt ggcagcggcg ccaccaactt cagcctgctt 960
aaacaagctg gcgacgtgga ggagaatcct ggcccaatga agaaactgct ggccatgatc 1020
ctgtggctgc agctggatag actgagcggc gagctgaagg tggaacagaa ccctctgttc 1080
ctgagcatgc aagagggcaa gaactacacc atctactgca actacagcac caccagcgac 1140
cggctgtact ggtacagaca ggatccaggc aagagcctgg aaagcctgtt cgtgctgctg 1200
agcaatggcg ccgtgaaaca agagggtaga ctgatggcca gcctggacac caaggccaga 1260
ctgagcacac tgcacattac agccgccgtg cacgatctga gcgccaccta tttttgtgcc 1320
ggctactctg gcgccggaag ctaccagctg acatttggca agggcaccaa gctgagcgtg 1380
atccccaata tccagaatcc ggaccctgcc gtctaccagc tgagggactc aaagagctcc 1440
gataaaagcg tgtgcctgtt caccgacttt gattctcaga caaacgtcag ccagtccaag 1500
gacagtgacg tgtacattac agacaaatgc gtgctggata tgcgctccat ggacttcaag 1560
tctaacagtg cagtggcctg gtccaacaag tctgatttcg cttgcgcaaa cgcctttaac 1620
aattcaatca ttccagagga taccttcttt ccaagccccg aatctagttg tgacgtgaag 1680
ctggtcgaga aatctttcga aacagatact aacctgaatt ttcagaatct gagcgtgatc 1740
ggcttccgca ttctgctgct gaaagtggct gggtttaatc tgctgatgac tctgcggctg 1800
tggtcttcct aa 1812
<210> 455
<211> 1839
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 38 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 455
atgggacctg gactgctttg ttgggtgctg ctgtgtctgc tcggagccgg atctgtggaa 60
acaggcgtga cacagagccc cacacacctg atcaagacca gaggccagca agtgaccctg 120
agatgcagct ctcagagcgg ccacaatacc gtgtcctggt atcagcaggc cctcggacag 180
ggccctcagt tcatcttcca gtactacaga gaggaagaga acggcagagg caacttccca 240
cctagattca gcggcctgca gttccccaac tacagcagcg agctgaacgt gaacgccctg 300
gaactggatg acagcgccct gtacctgtgt gcctcttctc ttgtggctgg cggcgagaca 360
cagtactttg gccccggaac aagactgctg gtgctggaag atctgaagaa cgtcttccca 420
cctgaggtgg ccgtgttcga gccttctgag gccgagatca gccacacaca gaaagccaca 480
ctcgtgtgtc tggccaccgg cttctatccc gatcacgtgg aactgtcttg gtgggtcaac 540
ggcaaagagg tgcacagcgg cgtctgtacc gatcctcagc ctctgaaaga gcagcccgct 600
ctgaacgaca gcagatactg cctgagcagc agactgagag tgtccgccac cttctggcag 660
aaccccagaa accacttcag atgccaggtg cagttctacg gcctgagcga gaacgatgag 720
tggacccagg atagagccaa gcctgtgaca cagatcgtgt ctgccgaagc ctggggcaga 780
gccgattgtg gctttaccag cgagagctac cagcagggcg tgctgtctgc cacaatcctg 840
tacgagatcc tgctgggcaa agccactctg tacgccgtgc tggtgtctgc cctggtgctg 900
atggccatgg tcaagcggaa ggatagcaga ggcggaagcg gcgccacaaa cttctcactg 960
ctgaaacagg ctggcgacgt ggaggagaat cctggcccaa tggtcaagat gcctggcgct 1020
agacggcaga gcatcatgaa gagaattctg ggcgccctgc tgggcctgct gtctgcccaa 1080
gtttgttgtg tgcggggcat ccaggtggaa cagagccctc ctgatctgat cctgcaagag 1140
ggcgccaaca gcaccctgag atgcaacttc agcgacagcg tgaacaacct gcagtggttc 1200
caccagaatc cttggggcca gctgatcaac ctgttctaca tccccagcgg caccaagcag 1260
aacggcagac tgtctgctac caccgtggcc accgagagat acagcctgct gtacatcagc 1320
agcagccaga ccacagacag cggcgtgtac ttttgcgccg tgggcttcaa cgacatgaga 1380
ttcggagccg gcaccagact gaccgtgaag cctaatatcc agaatccgga ccctgccgtc 1440
taccagctga gggactcaaa gagctccgat aaaagcgtgt gcctgttcac cgactttgat 1500
tctcagacaa acgtcagcca gtccaaggac agtgacgtgt acattacaga caaatgcgtg 1560
ctggatatgc gctccatgga cttcaagtct aacagtgcag tggcctggtc caacaagtct 1620
gatttcgctt gcgcaaacgc ctttaacaat tcaatcattc cagaggatac cttctttcca 1680
agccccgaat ctagttgtga cgtgaagctg gtcgagaaat ctttcgaaac agatactaac 1740
ctgaattttc agaatctgag cgtgatcggc ttccgcattc tgctgctgaa agtggctggg 1800
tttaatctgc tgatgactct gcggctgtgg tcttcctaa 1839
<210> 456
<211> 1779
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 39 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 456
atggatactt ggcttgtgtg ctgggccatc ttcagcctgc tgaaggccgg actgacagag 60
cccgaagtga cacagacacc cagccaccaa gtgacccaga tgggccaaga agtgatcctg 120
cgctgcgtgc ccatcagcaa ccacctgtac ttctactggt acagacagat cctgggccag 180
aaagtggaat tcctggtgtc cttctacaac aacgagatca gcgagaagtc cgagatcttc 240
gacgaccagt tcagcgtgga aagacccgac ggcagcaact tcaccctgaa gatcagaagc 300
accaagctcg aggacagcgc catgtacttt tgcgccagca cacccagaga cagaggcaaa 360
gaggccttct tcggccaagg caccagactg acagtggtcg aggacctgaa taaggtgttc 420
cctccagagg tggccgtgtt cgagccttct gaggccgaga tcagccacac acagaaagcc 480
acactcgtgt gcctggccac cggctttttt cccgatcacg tggaactgtc ttggtgggtc 540
aacggcaaag aggtgcacag cggcgtctgt accgatcctc agcctctgaa agagcagccc 600
gctctgaacg acagcagata ctgcctgagc agcagactga gagtgtccgc caccttctgg 660
cagaacccca gaaaccactt cagatgccag gtgcagttct acggcctgag cgagaacgat 720
gagtggaccc aggatagagc caagcctgtg acacagatcg tgtctgccga agcctggggc 780
agagccgatt gtggctttac cagcgtgtcc tatcagcagg gcgtgctgtc tgccaccatc 840
ctgtatgaga tcctgctggg caaagccact ctgtacgccg tgctggtgtc tgccctggtg 900
ctgatggcca tggtcaagag aaaggacttt ggcagcggcg ccaccaactt cagcctgctt 960
aaacaagctg gcgacgtgga ggagaatcct ggcccaatgc agctgacatg ggtttccggc 1020
cagcagctga atcagagccc tcagagcatg ttcatccaag aaggcgagga cgtttccatg 1080
aactgcacca gcagcagcat cttcaacacc tggctgtggt acaagcaaga gcctggcgaa 1140
ggacccgtgc tgctgatcgc tctttacaag gccggcgagc tgaccagcaa cggcagactg 1200
acagcccagt tcggcattac ccggaaggac agcttcctga acatcagcgc cagcattccc 1260
agcgacgtgg gcatctattt ctgcgccggc tacagcagct ccaacgacta caagctgagc 1320
ttcggagccg gcaccaccgt gacagttaga gccaatatcc agaatccgga ccctgccgtc 1380
taccagctga gggactcaaa gagctccgat aaaagcgtgt gcctgttcac cgactttgat 1440
tctcagacaa acgtcagcca gtccaaggac agtgacgtgt acattacaga caaatgcgtg 1500
ctggatatgc gctccatgga cttcaagtct aacagtgcag tggcctggtc caacaagtct 1560
gatttcgctt gcgcaaacgc ctttaacaat tcaatcattc cagaggatac cttctttcca 1620
agccccgaat ctagttgtga cgtgaagctg gtcgagaaat ctttcgaaac agatactaac 1680
ctgaattttc agaatctgag cgtgatcggc ttccgcattc tgctgctgaa agtggctggg 1740
tttaatctgc tgatgactct gcggctgtgg tcttcctaa 1779
<210> 457
<211> 1917
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 40 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 457
atggatactt ggcttgtgtg ctgggccatc ttcagcctgc tgaaggccgg actgacagag 60
cccgaagtga cacagacacc cagccaccaa gtgacccaga tgggccaaga agtgatcctg 120
cgctgcgtgc ccatcagcaa ccacctgtac ttctactggt acagacagat cctgggccag 180
aaagtggaat tcctggtgtc cttctacaac aacgagatca gcgagaagtc cgagatcttc 240
gacgaccagt tcagcgtgga aagacccgac ggcagcaact tcaccctgaa gatcagaagc 300
accaagctcg aggacagcgc catgtacttt tgcgccatca ccgccagaag cagctacgag 360
cagtatttcg gccctggcac cagactgacc gtgacagaag atctgaagaa cgtcttccca 420
cctgaggtgg ccgtgttcga gccttctgag gccgagatca gccacacaca gaaagccaca 480
ctcgtgtgtc tggccaccgg cttctatccc gatcacgtgg aactgtcttg gtgggtcaac 540
ggcaaagagg tgcacagcgg cgtctgtacc gatcctcagc ctctgaaaga gcagcccgct 600
ctgaacgaca gcagatactg cctgagcagc agactgagag tgtccgccac cttctggcag 660
aaccccagaa accacttcag atgccaggtg cagttctacg gcctgagcga gaacgatgag 720
tggacccagg atagagccaa gcctgtgaca cagatcgtgt ctgccgaagc ctggggcaga 780
gccgattgtg gctttaccag cgagagctac cagcagggcg tgctgtctgc cacaatcctg 840
tacgagatcc tgctgggcaa agccactctg tacgccgtgc tggtgtctgc cctggtgctg 900
atggccatgg tcaagcggaa ggatagcaga ggcggaagcg gcgccacaaa cttctcactg 960
ctgaaacagg ctggcgacgt ggaggagaat cctggcccaa tgcacaccag caccttccag 1020
aacagacccc agctgttcct gctgatctgg aagaaactgg tgcccggcaa tcccttcagg 1080
cggtcttgga tgaagcgcga gagagagatg ctgctgatca cctccatgct ggtgctgtgg 1140
atgcagctga gccaagtgaa cggccagcaa gtgatgcaga tccctcagta ccagcacgtg 1200
caagaaggcg aggacttcac cacctactgc aacagcagca ccacactgag caacatccag 1260
tggtacaagc agcggcctgg cggacaccct gtgtttctga tccagctggt caagtccggc 1320
gaagtgaaga agcagaagcg gctgaccttc cagttcggcg aggccaagaa gaacagcagc 1380
ctgcacatca ccgccacaca gaccaccgat gtgggcacct acttttgcgc cggcagaaac 1440
aacttcaaca agttctactt cggcagcggc accaagctga acgtgaagcc caatatccag 1500
aatccggacc ctgccgtcta ccagctgagg gactcaaaga gctccgataa aagcgtgtgc 1560
ctgttcaccg actttgattc tcagacaaac gtcagccagt ccaaggacag tgacgtgtac 1620
attacagaca aatgcgtgct ggatatgcgc tccatggact tcaagtctaa cagtgcagtg 1680
gcctggtcca acaagtctga tttcgcttgc gcaaacgcct ttaacaattc aatcattcca 1740
gaggatacct tctttccaag ccccgaatct agttgtgacg tgaagctggt cgagaaatct 1800
ttcgaaacag atactaacct gaattttcag aatctgagcg tgatcggctt ccgcattctg 1860
ctgctgaaag tggctgggtt taatctgctg atgactctgc ggctgtggtc ttcctaa 1917
<210> 458
<211> 1833
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 41 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 458
atggatactt ggcttgtgtg ctgggccatc ttcagcctgc tgaaggccgg actgacagag 60
cccgaagtga cacagacacc cagccaccaa gtgacccaga tgggccaaga agtgatcctg 120
cgctgcgtgc ccatcagcaa ccacctgtac ttctactggt acagacagat cctgggccag 180
aaagtggaat tcctggtgtc cttctacaac aacgagatca gcgagaagtc cgagatcttc 240
gacgaccagt tcagcgtgga aagacccgac ggcagcaact tcaccctgaa gatcagaagc 300
accaagctcg aggacagcgc catgtacttt tgcgccagca atccccggga cagagtgtcc 360
tacgagcagt actttggccc tggcaccaga ctgaccgtga cagaagatct gaagaacgtc 420
ttcccacctg aggtggccgt gttcgagcct tctgaggccg agatcagcca cacacagaaa 480
gccacactcg tgtgtctggc caccggcttc tatcccgatc acgtggaact gtcttggtgg 540
gtcaacggca aagaggtgca cagcggcgtc tgtaccgatc ctcagcctct gaaagagcag 600
cccgctctga acgacagcag atactgcctg agcagcagac tgagagtgtc cgccaccttc 660
tggcagaacc ccagaaacca cttcagatgc caggtgcagt tctacggcct gagcgagaac 720
gatgagtgga cccaggatag agccaagcct gtgacacaga tcgtgtctgc cgaagcctgg 780
ggcagagccg attgtggctt taccagcgag agctaccagc agggcgtgct gtctgccaca 840
atcctgtacg agatcctgct gggcaaagcc actctgtacg ccgtgctggt gtctgccctg 900
gtgctgatgg ccatggtcaa gcggaaggat agcagaggcg gaagcggcgc cacaaacttc 960
tcactgctga aacaggctgg cgacgtggag gagaatcctg gcccaatgat gatcagcctg 1020
cgggtgctgc tggtcatcct gtggctgcaa ctgagctggg tctggtccca gcggaaagag 1080
gtggaacagg accccggacc tttcaatgtg cctgaaggcg ccaccgtggc cttcaactgc 1140
acctacagca atagcgccag ccagagcttc ttctggtaca gacaggactg ccggaaagaa 1200
cccaagctgc tgatgagcgt gtacagcagc ggcaacgagg acggcagatt cacagcccag 1260
ctgaacagag ccagccagta catctccctg ctgatccggg atagcaagct gagcgatagc 1320
gccacctacc tgtgcgtggt cacatttgct ctgaccggcg gcttcaagac catcttcgga 1380
gccggcacca gactgttcgt gaaggccaat atccagaatc cggaccctgc cgtctaccag 1440
ctgagggact caaagagctc cgataaaagc gtgtgcctgt tcaccgactt tgattctcag 1500
acaaacgtca gccagtccaa ggacagtgac gtgtacatta cagacaaatg cgtgctggat 1560
atgcgctcca tggacttcaa gtctaacagt gcagtggcct ggtccaacaa gtctgatttc 1620
gcttgcgcaa acgcctttaa caattcaatc attccagagg ataccttctt tccaagcccc 1680
gaatctagtt gtgacgtgaa gctggtcgag aaatctttcg aaacagatac taacctgaat 1740
tttcagaatc tgagcgtgat cggcttccgc attctgctgc tgaaagtggc tgggtttaat 1800
ctgctgatga ctctgcggct gtggtcttcc taa 1833
<210> 459
<211> 1917
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 42 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 459
atggatactt ggcttgtgtg ctgggccatc ttcagcctgc tgaaggccgg actgacagag 60
cccgaagtga cacagacacc cagccaccaa gtgacccaga tgggccaaga agtgatcctg 120
cgctgcgtgc ccatcagcaa ccacctgtac ttctactggt acagacagat cctgggccag 180
aaagtggaat tcctggtgtc cttctacaac aacgagatca gcgagaagtc cgagatcttc 240
gacgaccagt tcagcgtgga aagacccgac ggcagcaact tcaccctgaa gatcagaagc 300
accaagctcg aggacagcgc catgtacttt tgcgccaaga ccagcagaag cagctacgag 360
cagtacttcg gccctggcac cagactgaca gtgacagaag atctgaagaa cgtcttccca 420
cctgaggtgg ccgtgttcga gccttctgag gccgagatca gccacacaca gaaagccaca 480
ctcgtgtgtc tggccaccgg cttctatccc gatcacgtgg aactgtcttg gtgggtcaac 540
ggcaaagagg tgcacagcgg cgtctgtacc gatcctcagc ctctgaaaga gcagcccgct 600
ctgaacgaca gcagatactg cctgagcagc agactgagag tgtccgccac cttctggcag 660
aaccccagaa accacttcag atgccaggtg cagttctacg gcctgagcga gaacgatgag 720
tggacccagg atagagccaa gcctgtgaca cagatcgtgt ctgccgaagc ctggggcaga 780
gccgattgtg gctttaccag cgagagctac cagcagggcg tgctgtctgc cacaatcctg 840
tacgagatcc tgctgggcaa agccactctg tacgccgtgc tggtgtctgc cctggtgctg 900
atggccatgg tcaagcggaa ggatagcaga ggcggaagcg gcgccacaaa cttctcactg 960
ctgaaacagg ctggcgacgt ggaggagaat cctggcccaa tgcacaccag caccttccag 1020
aacagacccc agctgttcct gctgatctgg aagaaactgg tgcccggcaa tcccttcagg 1080
cggtcttgga tgaagcgcga gagagagatg ctgctgatca cctccatgct ggtgctgtgg 1140
atgcagctga gccaagtgaa cggccagcaa gtgatgcaga tccctcagta ccagcacgtg 1200
caagaaggcg aggacttcac cacctactgc aacagcagca ccacactgag caacatccag 1260
tggtacaagc agcggcctgg cggacaccct gtgtttctga tccagctggt caagtccggc 1320
gaagtgaaga agcagaagcg gctgaccttc cagttcggcg aggccaagaa gaacagcagc 1380
ctgcacatca ccgccacaca gaccaccgat gtgggcacct acttttgcgc cggacctgac 1440
aacttcaaca agttctactt cggcagcggc accaagctga acgtgaagcc caatatccag 1500
aatccggacc ctgccgtcta ccagctgagg gactcaaaga gctccgataa aagcgtgtgc 1560
ctgttcaccg actttgattc tcagacaaac gtcagccagt ccaaggacag tgacgtgtac 1620
attacagaca aatgcgtgct ggatatgcgc tccatggact tcaagtctaa cagtgcagtg 1680
gcctggtcca acaagtctga tttcgcttgc gcaaacgcct ttaacaattc aatcattcca 1740
gaggatacct tctttccaag ccccgaatct agttgtgacg tgaagctggt cgagaaatct 1800
ttcgaaacag atactaacct gaattttcag aatctgagcg tgatcggctt ccgcattctg 1860
ctgctgaaag tggctgggtt taatctgctg atgactctgc ggctgtggtc ttcctaa 1917
<210> 460
<211> 1830
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 43 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 460
atggatactt ggcttgtgtg ctgggccatc ttcagcctgc tgaaggccgg actgacagag 60
cccgaagtga cacagacacc cagccaccaa gtgacccaga tgggccaaga agtgatcctg 120
cgctgcgtgc ccatcagcaa ccacctgtac ttctactggt acagacagat cctgggccag 180
aaagtggaat tcctggtgtc cttctacaac aacgagatca gcgagaagtc cgagatcttc 240
gacgaccagt tcagcgtgga aagacccgac ggcagcaact tcaccctgaa gatcagaagc 300
accaagctcg aggacagcgc catgtacttt tgcgccagca cacccagaga cagctacgag 360
cagtatttcg gccctggcac cagactgacc gtgacagaag atctgaagaa cgtcttccca 420
cctgaggtgg ccgtgttcga gccttctgag gccgagatca gccacacaca gaaagccaca 480
ctcgtgtgtc tggccaccgg cttctatccc gatcacgtgg aactgtcttg gtgggtcaac 540
ggcaaagagg tgcacagcgg cgtctgtacc gatcctcagc ctctgaaaga gcagcccgct 600
ctgaacgaca gcagatactg cctgagcagc agactgagag tgtccgccac cttctggcag 660
aaccccagaa accacttcag atgccaggtg cagttctacg gcctgagcga gaacgatgag 720
tggacccagg atagagccaa gcctgtgaca cagatcgtgt ctgccgaagc ctggggcaga 780
gccgattgtg gctttaccag cgagagctac cagcagggcg tgctgtctgc cacaatcctg 840
tacgagatcc tgctgggcaa agccactctg tacgccgtgc tggtgtctgc cctggtgctg 900
atggccatgg tcaagcggaa ggatagcaga ggcggaagcg gcgccacaaa cttctcactg 960
ctgaaacagg ctggcgacgt ggaggagaat cctggcccaa tgatgaagtc cctgcgggtg 1020
ctgctggtca tcctgtggct gcaactgagc tgggtctggt cccagcagaa agaggtggaa 1080
cagaacagcg gccctctgtc tgttcctgaa ggcgctatcg ccagcctgaa ctgcacctac 1140
agcgatagag gcagccagag cttcttctgg tacagacagt acagcggcaa gagccccgag 1200
ctgatcatgt tcatctacag caacggcgac aaagaggacg gccggtttac agcccagctg 1260
aacaaggcca gccagtacgt gtccctgctg atcagagata gccagcctag cgacagcgcc 1320
acctatctgt gtgccgtgaa tgtgcctacc agcggcacct acaagtacat cttcggcacc 1380
ggcaccagac tgaaggtgct ggctaatatc cagaatccgg accctgccgt ctaccagctg 1440
agggactcaa agagctccga taaaagcgtg tgcctgttca ccgactttga ttctcagaca 1500
aacgtcagcc agtccaagga cagtgacgtg tacattacag acaaatgcgt gctggatatg 1560
cgctccatgg acttcaagtc taacagtgca gtggcctggt ccaacaagtc tgatttcgct 1620
tgcgcaaacg cctttaacaa ttcaatcatt ccagaggata ccttctttcc aagccccgaa 1680
tctagttgtg acgtgaagct ggtcgagaaa tctttcgaaa cagatactaa cctgaatttt 1740
cagaatctga gcgtgatcgg cttccgcatt ctgctgctga aagtggctgg gtttaatctg 1800
ctgatgactc tgcggctgtg gtcttcctaa 1830
<210> 461
<211> 1851
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 44 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 461
atgggcttta gactgctgtg ctgcgtggcc ttctgtctgc ttggagctgg ccctgtggat 60
agcggcgtta cccagacacc taagcacctg atcacagcca caggccagcg cgtgaccctg 120
agatgttctc ctagaagcgg cgacctgagc gtgtactggt atcagcagtc tctggaccag 180
ggcctgcagt tcctgatcca gtactacaac ggcgaggaaa gagccaaggg caacatcctg 240
gaacggttca gcgcccagca gttcccagat ctgcacagcg agctgaacct gagcagcctg 300
gaactgggag atagcgccct gtacttctgt gccagcagcg gcacacccga cacacagtat 360
tttggccctg gcaccagact gaccgtgctg gaagatctga agaacgtctt cccacctgag 420
gtggccgtgt tcgagccttc tgaggccgag atcagccaca cacagaaagc cacactcgtg 480
tgtctggcca ccggcttcta tcccgatcac gtggaactgt cttggtgggt caacggcaaa 540
gaggtgcaca gcggcgtctg taccgatcct cagcctctga aagagcagcc cgctctgaac 600
gacagcagat actgcctgag cagcagactg agagtgtccg ccaccttctg gcagaacccc 660
agaaaccact tcagatgcca ggtgcagttc tacggcctga gcgagaacga tgagtggacc 720
caggatagag ccaagcctgt gacacagatc gtgtctgccg aagcctgggg cagagccgat 780
tgtggcttta ccagcgagag ctaccagcag ggcgtgctgt ctgccacaat cctgtacgag 840
atcctgctgg gcaaagccac tctgtacgcc gtgctggtgt ctgccctggt gctgatggcc 900
atggtcaagc ggaaggatag cagaggcgga agcggcgcca caaacttctc actgctgaaa 960
caggctggcg acgtggagga gaatcctggc ccaatggctc aagagctggg catgcagtgt 1020
caggccagag gaatcctgca gcagatgtgg ggagtgttcc tgctgtacgt gtccatgaag 1080
atgggcggca ccaccggcca gaacatcgat cagcctacag agatgaccgc caccgagggc 1140
gccatcgtgc agatcaattg cacctaccag accagcggct tcaacggcct gttctggtat 1200
cagcagcatg ccggcgaggc ccctaccttc ctgagctaca atgtgctgga cggcctggaa 1260
gagaagggca gattcagcag cttcctgtcc agaagcaagg gctacagcta cctgctgctg 1320
aaagaactgc agatgaagga cagcgcctcc tacctgtgtg cccagtactc tggcggctac 1380
cagaaagtga cctttggcac cggcaccaag ctgcaagtga tccccaatat ccagaatccg 1440
gaccctgccg tctaccagct gagggactca aagagctccg ataaaagcgt gtgcctgttc 1500
accgactttg attctcagac aaacgtcagc cagtccaagg acagtgacgt gtacattaca 1560
gacaaatgcg tgctggatat gcgctccatg gacttcaagt ctaacagtgc agtggcctgg 1620
tccaacaagt ctgatttcgc ttgcgcaaac gcctttaaca attcaatcat tccagaggat 1680
accttctttc caagccccga atctagttgt gacgtgaagc tggtcgagaa atctttcgaa 1740
acagatacta acctgaattt tcagaatctg agcgtgatcg gcttccgcat tctgctgctg 1800
aaagtggctg ggtttaatct gctgatgact ctgcggctgt ggtcttccta a 1851
<210> 462
<211> 1929
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 45 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 462
atgggaacca gactgctgtt ctgggtcgcc ttctgtctgc tgggcgccta tcatacaggt 60
gccggtgtct ctcagagccc cagcaacaaa gtgaccgaga agggcaaaga cgtggaactg 120
agatgcgacc ccatcagcgg acacacagcc ctgtactggt acagacagag actcggccag 180
ggcctcgagt tcctgatcta cttccaaggc aacagcgccc ctgacaagag cggcctgcct 240
agcgatagat tcagcgccga gagaacaggc gagagcgtgt ccacactgac catccagaga 300
acccagcaag aggacagcgc cgtgtacctg tgtgccagct ctctgtacct gggaaccacc 360
ggcgagctgt tctttggcga gggctctaga ctgaccgtgc tggaagatct gaagaacgtc 420
ttcccacctg aggtggccgt gttcgagcct tctgaggccg agatcagcca cacacagaaa 480
gccacactcg tgtgtctggc caccggcttc tatcccgatc acgtggaact gtcttggtgg 540
gtcaacggca aagaggtgca cagcggcgtc tgtaccgatc ctcagcctct gaaagagcag 600
cccgctctga acgacagcag atactgcctg agcagcagac tgagagtgtc cgccaccttc 660
tggcagaacc ccagaaacca cttcagatgc caggtgcagt tctacggcct gagcgagaac 720
gatgagtgga cccaggatag agccaagcct gtgacacaga tcgtgtctgc cgaagcctgg 780
ggcagagccg attgtggctt taccagcgag agctaccagc agggcgtgct gtctgccaca 840
atcctgtacg agatcctgct gggcaaagcc actctgtacg ccgtgctggt gtctgccctg 900
gtgctgatgg ccatggtcaa gcggaaggat agcagaggcg gaagcggcgc cacaaacttc 960
tcactgctga aacaggctgg cgacgtggag gagaatcctg gcccaatgca caccagcacc 1020
ttccagaaca gaccccagct gttcctgctg atctggaaga aactggtgcc cggcaatccc 1080
ttcaggcggt cttggatgaa gcgcgagaga gagatgctgc tgatcacctc catgctggtg 1140
ctgtggatgc agctgagcca agtgaacggc cagcaagtga tgcagatccc tcagtaccag 1200
cacgtgcaag aaggcgagga cttcaccacc tactgcaaca gcagcaccac actgagcaac 1260
atccagtggt acaagcagcg gcctggcgga caccctgtgt ttctgatcca gctggtcaag 1320
tccggcgaag tgaagaagca gaagcggctg accttccagt tcggcgaggc caagaagaac 1380
agcagcctgc acatcaccgc cacacagacc accgatgtgg gcacctattt ttgcgccgga 1440
tcttctggcg ccggaagcta ccagctgaca tttggcaagg gcaccaagct gagcgtgatc 1500
cccaatatcc agaatccgga ccctgccgtc taccagctga gggactcaaa gagctccgat 1560
aaaagcgtgt gcctgttcac cgactttgat tctcagacaa acgtcagcca gtccaaggac 1620
agtgacgtgt acattacaga caaatgcgtg ctggatatgc gctccatgga cttcaagtct 1680
aacagtgcag tggcctggtc caacaagtct gatttcgctt gcgcaaacgc ctttaacaat 1740
tcaatcattc cagaggatac cttctttcca agccccgaat ctagttgtga cgtgaagctg 1800
gtcgagaaat ctttcgaaac agatactaac ctgaattttc agaatctgag cgtgatcggc 1860
ttccgcattc tgctgctgaa agtggctggg tttaatctgc tgatgactct gcggctgtgg 1920
tcttcctaa 1929
<210> 463
<211> 1830
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 46 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 463
atgggaacaa gactgctgtg ttgggccgct ctgtgtctgc tgggagctga tcatacaggc 60
gccggtgtca gccagacacc tagcaacaaa gtgaccgaga agggcaaata cgtggaactg 120
agatgcgacc ccatcagcgg acacacagcc ctgtactggt acagacagtc tctcggccag 180
ggacctgagt tcctgatcta cttccaaggc accggcgctg ccgatgatag cggcctgcct 240
aacgatagat tcttcgccgt cagacccgag ggcagcgtgt ccacactgaa gatccagaga 300
accgagaggg gcgacagcgc cgtgtatctg tgtgccagct ctctgtatct cggcggcagc 360
gagacacagt actttggccc tggtacacgg ctgctggttc tggaagatct gaagaacgtc 420
ttcccacctg aggtggccgt gttcgagcct tctgaggccg agatcagcca cacacagaaa 480
gccacactcg tgtgtctggc caccggcttc tatcccgatc acgtggaact gtcttggtgg 540
gtcaacggca aagaggtgca cagcggcgtc tgtaccgatc ctcagcctct gaaagagcag 600
cccgctctga acgacagcag atactgcctg agcagcagac tgagagtgtc cgccaccttc 660
tggcagaacc ccagaaacca cttcagatgc caggtgcagt tctacggcct gagcgagaac 720
gatgagtgga cccaggatag agccaagcct gtgacacaga tcgtgtctgc cgaagcctgg 780
ggcagagccg attgtggctt taccagcgag agctaccagc agggcgtgct gtctgccaca 840
atcctgtacg agatcctgct gggcaaagcc actctgtacg ccgtgctggt gtctgccctg 900
gtgctgatgg ccatggtcaa gcggaaggat agcagaggcg gaagcggcgc cacaaacttc 960
tcactgctga aacaggctgg cgacgtggag gagaatcctg gcccaatgct gctgcttctg 1020
gtgcccgcct tccaagtgat ctttaccctc ggcggaacac gggcccagtc tgtgacacag 1080
ctggattctc aggtgcccgt gtttgaggaa gcccctgtcg agctgagatg caactacagc 1140
agcagcgtgt ccgtgtacct gttttggtac gtgcagtacc ccaaccaggg cctgcagctg 1200
ctgctcaagt atctgagcgg cagcacactg gtcaagggca tcaatggctt cgaggccgag 1260
ttcaacaaga gccagaccag cttccatctg cggaagccca gcgtgcacat cagcgatacc 1320
gccgagtact tctgcgccgt gtctcctagc agcggcacct acaagtacat cttcggcacc 1380
ggcaccagac tgaaggtgct ggctaatatc cagaatccgg accctgccgt ctaccagctg 1440
agggactcaa agagctccga taaaagcgtg tgcctgttca ccgactttga ttctcagaca 1500
aacgtcagcc agtccaagga cagtgacgtg tacattacag acaaatgcgt gctggatatg 1560
cgctccatgg acttcaagtc taacagtgca gtggcctggt ccaacaagtc tgatttcgct 1620
tgcgcaaacg cctttaacaa ttcaatcatt ccagaggata ccttctttcc aagccccgaa 1680
tctagttgtg acgtgaagct ggtcgagaaa tctttcgaaa cagatactaa cctgaatttt 1740
cagaatctga gcgtgatcgg cttccgcatt ctgctgctga aagtggctgg gtttaatctg 1800
ctgatgactc tgcggctgtg gtcttcctaa 1830
<210> 464
<211> 1830
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 47 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 464
atggatactt ggcttgtgtg ctgggccatc ttcagcctgc tgaaggccgg actgacagag 60
cccgaagtga cacagacacc cagccaccaa gtgacccaga tgggccaaga agtgatcctg 120
cgctgcgtgc ccatcagcaa ccacctgtac ttctactggt acagacagat cctgggccag 180
aaagtggaat tcctggtgtc cttctacaac aacgagatca gcgagaagtc cgagatcttc 240
gacgaccagt tcagcgtgga aagacccgac ggcagcaact tcaccctgaa gatcagaagc 300
accaagctcg aggacagcgc catgtacttt tgcgccatga ccggcagaac cacctacgag 360
cagtatttcg gccctggcac cagactgacc gtgacagaag atctgaagaa cgtcttccca 420
cctgaggtgg ccgtgttcga gccttctgag gccgagatca gccacacaca gaaagccaca 480
ctcgtgtgtc tggccaccgg cttctatccc gatcacgtgg aactgtcttg gtgggtcaac 540
ggcaaagagg tgcacagcgg cgtctgtacc gatcctcagc ctctgaaaga gcagcccgct 600
ctgaacgaca gcagatactg cctgagcagc agactgagag tgtccgccac cttctggcag 660
aaccccagaa accacttcag atgccaggtg cagttctacg gcctgagcga gaacgatgag 720
tggacccagg atagagccaa gcctgtgaca cagatcgtgt ctgccgaagc ctggggcaga 780
gccgattgtg gctttaccag cgagagctac cagcagggcg tgctgtctgc cacaatcctg 840
tacgagatcc tgctgggcaa agccactctg tacgccgtgc tggtgtctgc cctggtgctg 900
atggccatgg tcaagcggaa ggatagcaga ggcggaagcg gcgccacaaa cttctcactg 960
ctgaaacagg ctggcgacgt ggaggagaat cctggcccaa tgatgaagtc cctgcgggtg 1020
ctgctggtca tcctgtggct gcaactgagc tgggtctggt cccagcagaa agaggtggaa 1080
cagaacagcg gccctctgtc tgttcctgaa ggcgctatcg ccagcctgaa ctgcacctac 1140
agcgatagag gcagccagag cttcttctgg tacagacagt acagcggcaa gagccccgag 1200
ctgatcatgt tcatctacag caacggcgac aaagaggacg gccggtttac agcccagctg 1260
aacaaggcca gccagtacgt gtccctgctg atcagagata gccagcctag cgacagcgcc 1320
acctatctgt gtgccgtgaa tctgctgagc ggcagcgcta gacagctgac atttggctct 1380
ggcacccagc tgaccgtgct gcctgatatc cagaatccgg accctgccgt ctaccagctg 1440
agggactcaa agagctccga taaaagcgtg tgcctgttca ccgactttga ttctcagaca 1500
aacgtcagcc agtccaagga cagtgacgtg tacattacag acaaatgcgt gctggatatg 1560
cgctccatgg acttcaagtc taacagtgca gtggcctggt ccaacaagtc tgatttcgct 1620
tgcgcaaacg cctttaacaa ttcaatcatt ccagaggata ccttctttcc aagccccgaa 1680
tctagttgtg acgtgaagct ggtcgagaaa tctttcgaaa cagatactaa cctgaatttt 1740
cagaatctga gcgtgatcgg cttccgcatt ctgctgctga aagtggctgg gtttaatctg 1800
ctgatgactc tgcggctgtg gtcttcctaa 1830
<210> 465
<211> 1824
<212> ДНК
<213> Homo sapiens
<220>
<221> iДНК
<222> (0)...(0)
<223> TCR 48 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 465
atggatactt ggcttgtgtg ctgggccatc ttcagcctgc tgaaggccgg actgacagag 60
cccgaagtga cacagacacc cagccaccaa gtgacccaga tgggccaaga agtgatcctg 120
cgctgcgtgc ccatcagcaa ccacctgtac ttctactggt acagacagat cctgggccag 180
aaagtggaat tcctggtgtc cttctacaac aacgagatca gcgagaagtc cgagatcttc 240
gacgaccagt tcagcgtgga aagacccgac ggcagcaact tcaccctgaa gatcagaagc 300
accaagctcg aggacagcgc catgtacttt tgcgccagca ccggcagagt gtcctacgag 360
cagtatttcg gccctggcac cagactgacc gtgacagaag atctgaagaa cgtcttccca 420
cctgaggtgg ccgtgttcga gccttctgag gccgagatca gccacacaca gaaagccaca 480
ctcgtgtgtc tggccaccgg cttctatccc gatcacgtgg aactgtcttg gtgggtcaac 540
ggcaaagagg tgcacagcgg cgtctgtacc gatcctcagc ctctgaaaga gcagcccgct 600
ctgaacgaca gcagatactg cctgagcagc agactgagag tgtccgccac cttctggcag 660
aaccccagaa accacttcag atgccaggtg cagttctacg gcctgagcga gaacgatgag 720
tggacccagg atagagccaa gcctgtgaca cagatcgtgt ctgccgaagc ctggggcaga 780
gccgattgtg gctttaccag cgagagctac cagcagggcg tgctgtctgc cacaatcctg 840
tacgagatcc tgctgggcaa agccactctg tacgccgtgc tggtgtctgc cctggtgctg 900
atggccatgg tcaagcggaa ggatagcaga ggcggaagcg gcgccacaaa cttctcactg 960
ctgaaacagg ctggcgacgt ggaggagaat cctggcccaa tgatgaagtc cctgcgggtg 1020
ctgctggtca tcctgtggct gcaactgagc tgggtctggt cccagcagaa agaggtggaa 1080
caggaccctg gacctctgtc tgttcctgag ggcgccatcg tgtccctgaa ctgcacctac 1140
agcaacagcg ccttccagta cttcatgtgg tacagacagt acagccggaa gggccccgag 1200
ctgctgatgt acacatacag cagcggcaac aaagaggacg gccggtttac agcccaggtg 1260
gacaagagca gcaagtacat ctccctgttc atccgggaca gccagcctag cgatagcgcc 1320
acatatctgt gcgccatgag aatccagggc gcccagaaac tggtgttcgg ccagggaacc 1380
agactgacca tcaatcccaa catccagaat ccggaccctg ccgtctacca gctgagggac 1440
tcaaagagct ccgataaaag cgtgtgcctg ttcaccgact ttgattctca gacaaacgtc 1500
agccagtcca aggacagtga cgtgtacatt acagacaaat gcgtgctgga tatgcgctcc 1560
atggacttca agtctaacag tgcagtggcc tggtccaaca agtctgattt cgcttgcgca 1620
aacgccttta acaattcaat cattccagag gataccttct ttccaagccc cgaatctagt 1680
tgtgacgtga agctggtcga gaaatctttc gaaacagata ctaacctgaa ttttcagaat 1740
ctgagcgtga tcggcttccg cattctgctg ctgaaagtgg ctgggtttaa tctgctgatg 1800
actctgcggc tgtggtcttc ctaa 1824
<210> 466
<211> 1830
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 49 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 466
atggatacct ggctcgtgtg ctgggccatc ttcagcctgc tgaaagccgg actgaccgag 60
cctgaagtga cccagacacc tagccaccaa gtgacacaga tgggccaaga agtgatcctg 120
cgctgcgtgc ccatcagcaa ccacctgtac ttctactggt acagacagat cctgggccag 180
aaagtggaat tcctggtgtc cttctacaac aacgagatca gcgagaagtc cgagatcttc 240
gacgaccagt tcagcgtgga aagacccgac ggcagcaact tcaccctgaa gatcagaagc 300
accaagctcg aggacagcgc catgtacttt tgcgccagca caccccggta cagctacgag 360
cagtatttcg gccctggcac cagactgacc gtgacagaag atctgaagaa cgtcttccct 420
cctgaagtgg ccgtgtttga gccttctgag gccgagatca gccacacaca gaaagccaca 480
ctcgtgtgtc tggccaccgg cttctatccc gatcacgtgg aactgtcttg gtgggtcaac 540
ggcaaagagg tgcacagcgg cgtctgtacc gatcctcagc ctctgaaaga gcagcccgct 600
ctgaacgaca gcagatactg cctgagcagc agactgagag tgtccgccac cttctggcag 660
aaccccagaa accacttcag atgccaggtg cagttctacg gcctgagcga gaacgatgag 720
tggacccagg atagagccaa gcctgtgaca cagatcgtgt ctgccgaagc ctggggcaga 780
gccgattgtg gctttaccag cgagagctac cagcagggcg tgctgtctgc cacaatcctg 840
tacgagatcc tgctgggcaa agccactctg tacgccgtgc tggtgtctgc cctggtgctg 900
atggccatgg tcaagcggaa ggatagcaga ggcggaagcg gcgccacaaa cttctcactg 960
ctgaaacagg ccggcgacgt ggaggagaat cctggcccaa tgatgaagtc cctgcgggtg 1020
ctgctggtca tcctgtggct gcaactgagc tgggtctggt cccagcagaa agaggtggaa 1080
cagaacagcg gccctctgtc tgttcctgaa ggcgctatcg ccagcctgaa ctgcacctac 1140
agcgatagag gcagccagag cttcttctgg tacagacagt acagcggcaa gagccccgag 1200
ctgatcatgt tcatctacag caacggcgac aaagaggacg gccggtttac agcccagctg 1260
aacaaggcca gccagtacgt gtccctgctg atcagagata gccagcctag cgacagcgcc 1320
acctatctgt gcgccgtgaa tatcggcacc agcggcacct acaagtacat cttcggcacc 1380
ggcaccagac tgaaggtgct ggctaatatc cagaatccgg accctgccgt ctaccagctg 1440
agggactcaa agagctccga taaaagcgtg tgcctgttca ccgactttga ttctcagaca 1500
aacgtcagcc agtccaagga cagtgacgtg tacattacag acaaatgcgt gctggatatg 1560
cgctccatgg acttcaagtc taacagtgca gtggcctggt ccaacaagtc tgatttcgct 1620
tgcgcaaacg cctttaacaa ttcaatcatt ccagaggata ccttctttcc aagccccgaa 1680
tctagttgtg acgtgaagct ggtcgagaaa tctttcgaaa cagatactaa cctgaatttt 1740
cagaatctga gcgtgatcgg cttccgcatt ctgctgctga aagtggctgg gtttaatctg 1800
ctgatgactc tgcggctgtg gtcttcctaa 1830
<210> 467
<211> 1815
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 50 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 467
atgggcttca gactgctgtg ctgcgtggcc ttttgtctgc ttggagccgg acctgtggat 60
agcggcgtta cccagacacc taagcacctg atcacagcca caggccagcg cgtgaccctg 120
agatgttctc ctagaagcgg cgacctgagc gtgtactggt atcagcagtc tctggaccag 180
ggcctgcagt tcctgatcca ctactacaac ggcgaggaaa gagccaaggg caacatcctg 240
gaacggttca gcgcccagca gttcccagat ctgcacagcg agctgaacct gagcagcctg 300
gaactgggag atagcgccct gtacttctgt gccagcagcg ccacaagaga tgcctacggc 360
tacacatttg gcagcggcac cagactgaca gtggtggagg acctgaataa ggtgttccct 420
cctgaagtgg ccgtgtttga gccttctgag gccgagatca gccacacaca gaaagccaca 480
ctcgtgtgcc tggccaccgg cttttttccc gatcacgtgg aactgtcttg gtgggtcaac 540
ggcaaagagg tgcacagcgg cgtctgtacc gatcctcagc ctctgaaaga gcagcccgct 600
ctgaacgaca gcagatactg cctgagcagc agactgagag tgtccgccac cttctggcag 660
aaccccagaa accacttcag atgccaggtg cagttctacg gcctgagcga gaacgatgag 720
tggacccagg atagagccaa gcctgtgaca cagatcgtgt ctgccgaagc ctggggcaga 780
gccgattgtg gctttaccag cgtgtcctat cagcagggcg tgctgtctgc caccatcctg 840
tatgagatcc tgctgggcaa agccactctg tacgccgtgc tggtgtctgc cctggtgctg 900
atggccatgg tcaagagaaa ggactttggc agcggcgcca ccaacttcag cctgcttaaa 960
caggcaggcg acgtggagga gaatcctggc ccaatgaaga ccttcgccgg cttcagcttc 1020
ctgttcctgt ggctgcagct ggactgcatg agcagaggcg aggatgtgga acagagcctg 1080
tttctgagcg tccgcgaggg cgatagcagc gtgatcaatt gcacctacac cgacagcagc 1140
agcacctacc tgtactggta caagcaagag cctggcgccg gactgcagct gctgacctac 1200
atcttcagca acatggacat gaagcaggac cagcggctga ccgtgctgct gaacaagaag 1260
gacaagcacc tgagcctgcg gatcgccgat acacagacag gcgacagcgc catctacttt 1320
tgcgccgagt ctcctccagg cacctacaag tacatcttcg gcaccggcac cagactgaag 1380
gtgctggcta atatccagaa tccggaccct gccgtctacc agctgaggga ctcaaagagc 1440
tccgataaaa gcgtgtgcct gttcaccgac tttgattctc agacaaacgt cagccagtcc 1500
aaggacagtg acgtgtacat tacagacaaa tgcgtgctgg atatgcgctc catggacttc 1560
aagtctaaca gtgcagtggc ctggtccaac aagtctgatt tcgcttgcgc aaacgccttt 1620
aacaattcaa tcattccaga ggataccttc tttccaagcc ccgaatctag ttgtgacgtg 1680
aagctggtcg agaaatcttt cgaaacagat actaacctga attttcagaa tctgagcgtg 1740
atcggcttcc gcattctgct gctgaaagtg gctgggttta atctgctgat gactctgcgg 1800
ctgtggtctt cctaa 1815
<210> 468
<211> 1833
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 51 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 468
atggatacct ggctcgtgtg ctgggccatc ttcagcctgc tgaaagccgg actgaccgag 60
cctgaagtga cccagacacc tagccaccaa gtgacacaga tgggccaaga agtgatcctg 120
cgctgcgtgc ccatcagcaa ccacctgtac ttctactggt acagacagat cctgggccag 180
aaagtggaat tcctggtgtc cttctacaac aacgagatca gcgagaagtc cgagatcttc 240
gacgaccagt tcagcgtgga aagacccgac ggcagcaact tcaccctgaa gatcagaagc 300
accaagctcg aggacagcgc catgtacttt tgcgccattg ccagccgggt gtcctacgag 360
cagtatttcg gccctggcac cagactgacc gtgacagaag atctgaagaa cgtcttccct 420
cctgaagtgg ccgtgtttga gccttctgag gccgagatca gccacacaca gaaagccaca 480
ctcgtgtgtc tggccaccgg cttctatccc gatcacgtgg aactgtcttg gtgggtcaac 540
ggcaaagagg tgcacagcgg cgtctgtacc gatcctcagc ctctgaaaga gcagcccgct 600
ctgaacgaca gcagatactg cctgagcagc agactgagag tgtccgccac cttctggcag 660
aaccccagaa accacttcag atgccaggtg cagttctacg gcctgagcga gaacgatgag 720
tggacccagg atagagccaa gcctgtgaca cagatcgtgt ctgccgaagc ctggggcaga 780
gccgattgtg gctttaccag cgagagctac cagcagggcg tgctgtctgc cacaatcctg 840
tacgagatcc tgctgggcaa agccactctg tacgccgtgc tggtgtctgc cctggtgctg 900
atggccatgg tcaagcggaa ggatagcaga ggcggaagcg gcgccacaaa cttctcactg 960
ctgaaacagg ccggcgacgt ggaggagaat cctggcccaa tgatgaagtc cctgcgggtg 1020
ctgctggtca tcctgtggct gcaactgagc tgggtctggt cccagcagaa agaggtggaa 1080
cagaacagcg gccctctgtc tgttcctgaa ggcgctatcg ccagcctgaa ctgcacctac 1140
agcgatagag gcagccagag cttcttctgg tacagacagt acagcggcaa gagccccgag 1200
ctgatcatga gcatctacag caacggcgac aaagaggacg gccggtttac agcccagctg 1260
aacaaggcca gccagtacgt gtccctgctg atcagagata gccagcctag cgacagcgcc 1320
acctatctgt gcgccgtgaa tatgagaggc ggcggaagca actacaagct gacctttggc 1380
aagggcaccc tgctgaccgt gaatcccaat atccagaatc cggaccctgc cgtctaccag 1440
ctgagggact caaagagctc cgataaaagc gtgtgcctgt tcaccgactt tgattctcag 1500
acaaacgtca gccagtccaa ggacagtgac gtgtacatta cagacaaatg cgtgctggat 1560
atgcgctcca tggacttcaa gtctaacagt gcagtggcct ggtccaacaa gtctgatttc 1620
gcttgcgcaa acgcctttaa caattcaatc attccagagg ataccttctt tccaagcccc 1680
gaatctagtt gtgacgtgaa gctggtcgag aaatctttcg aaacagatac taacctgaat 1740
tttcagaatc tgagcgtgat cggcttccgc attctgctgc tgaaagtggc tgggtttaat 1800
ctgctgatga ctctgcggct gtggtcttcc taa 1833
<210> 469
<211> 1806
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 52 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 469
atgggcttca gactgctgtg ctgcgtggcc ttttgtctgc ttggagccgg acctgtggat 60
agcggcgtta cccagacacc taagcacctg atcacagcca caggccagcg cgtgaccctg 120
agatgttctc ctagaagcgg cgacctgagc gtgtactggt atcagcagtc tctggaccag 180
ggcctgcagt tcctgatcca gtactacaac ggcgaggaaa gagccaaggg caacatcctg 240
gaacggttca gcgcccagca gttcccagat ctgcacagcg agctgaacct gagcagcctg 300
gaactgggag atagcgccct gtacttctgt gccagctctg tgggcgatct gaacaacgag 360
cagttcttcg gccctggcac cagactgaca gttctggaag atctgaagaa cgtcttccct 420
cctgaagtgg ccgtgtttga gccttctgag gccgagatca gccacacaca gaaagccaca 480
ctcgtgtgtc tggccaccgg cttctatccc gatcacgtgg aactgtcttg gtgggtcaac 540
ggcaaagagg tgcacagcgg cgtctgtacc gatcctcagc ctctgaaaga gcagcccgct 600
ctgaacgaca gcagatactg cctgagcagc agactgagag tgtccgccac cttctggcag 660
aaccccagaa accacttcag atgccaggtg cagttctacg gcctgagcga gaacgatgag 720
tggacccagg atagagccaa gcctgtgaca cagatcgtgt ctgccgaagc ctggggcaga 780
gccgattgtg gctttaccag cgagagctac cagcagggcg tgctgtctgc cacaatcctg 840
tacgagatcc tgctgggcaa agccactctg tacgccgtgc tggtgtctgc cctggtgctg 900
atggccatgg tcaagcggaa ggatagcaga ggcggaagcg gcgccacaaa cttctcactg 960
ctgaaacagg ccggcgacgt ggaggagaat cctggcccaa tggtgctgaa gttcagcgtg 1020
tccatcctgt ggatccagct ggcctgggtt tccacacagc tgctggaaca gagccctcag 1080
ttcctgagca tccaagaggg cgagaacctg accgtgtact gcaacagcag cagcgtgttc 1140
agcagcctgc agtggtacag acaagagcct ggcgaaggac ctgtgctgct ggtcacagtt 1200
gtgacaggcg gcgaagtgaa gaagctgaag cggctgacct tccagttcgg cgacgccaga 1260
aaggatagct ccctgcacat taccgctgct cagccaggcg ataccggcct gtatctttgt 1320
gccggcgctc gggattacaa gctgtctttt ggcgccggaa ccaccgtgac agtgcgggct 1380
aatatccaga atccggaccc tgccgtctac cagctgaggg actcaaagag ctccgataaa 1440
agcgtgtgcc tgttcaccga ctttgattct cagacaaacg tcagccagtc caaggacagt 1500
gacgtgtaca ttacagacaa atgcgtgctg gatatgcgct ccatggactt caagtctaac 1560
agtgcagtgg cctggtccaa caagtctgat ttcgcttgcg caaacgcctt taacaattca 1620
atcattccag aggatacctt ctttccaagc cccgaatcta gttgtgacgt gaagctggtc 1680
gagaaatctt tcgaaacaga tactaacctg aattttcaga atctgagcgt gatcggcttc 1740
cgcattctgc tgctgaaagt ggctgggttt aatctgctga tgactctgcg gctgtggtct 1800
tcctaa 1806
<210> 470
<211> 1836
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 53 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 470
atgggcacaa gcctgctgtg ttgggtcgtg ctgggctttc tgggcacaga tcatacaggc 60
gccggtgtca gccagtctcc tcggtacaaa gtgaccaagc gcggacagga tgtggccctg 120
agatgcgatc ctatctctgg ccacgtgtcc ctgtactggt acagacaggc tctcggacag 180
ggccccgagt tcctgaccta cttcaattac gaggcccagc aggacaagag cggcctgcct 240
aacgatagat tcagcgccga aagacccgag ggcagcatca gcacactgac catccagaga 300
accgagcaga gggacagcgc catgtacaga tgtgcctcta gcggcagcgg caccagcggc 360
tacaatgagc agtttttcgg ccctggcacc agactgacag tgctggaaga tctgaagaac 420
gtcttccctc ctgaagtggc cgtgtttgag ccttctgagg ccgagatcag ccacacacag 480
aaagccacac tcgtgtgtct ggccaccggc ttctatcccg atcacgtgga actgtcttgg 540
tgggtcaacg gcaaagaggt gcacagcggc gtctgtaccg atcctcagcc tctgaaagag 600
cagcccgctc tgaacgacag cagatactgc ctgagcagca gactgagagt gtccgccacc 660
ttctggcaga accccagaaa ccacttcaga tgccaggtgc agttctacgg cctgagcgag 720
aacgatgagt ggacccagga tagagccaag cctgtgacac agatcgtgtc tgccgaagcc 780
tggggcagag ccgattgtgg ctttaccagc gagagctacc agcagggcgt gctgtctgcc 840
acaatcctgt acgagatcct gctgggcaaa gccactctgt acgccgtgct ggtgtctgcc 900
ctggtgctga tggccatggt caagcggaag gatagcagag gcggaagcgg cgccacaaac 960
ttctcactgc tgaaacaggc cggcgacgtg gaggagaatc ctggcccaat ggccagcgct 1020
cctatctcta tgctggccat gctgttcacc ctgagcggac tgagagcaca gtctgtggcc 1080
cagcctgagg accaagtgaa tgtggccgag ggcaatcccc tgaccgtgaa gtgtacctac 1140
agcgtgtccg gcaatcccta cctgttttgg tacgtgcagt accccaaccg gggcctgcag 1200
ttcctgctga agtacatcac cggcgacaac ctggtcaagg gcagctatgg attcgaggcc 1260
gagttcaaca agagccagac cagcttccac ctgaagaaac ccagcgctct ggtgtccgat 1320
agcgccctgt atttctgcgc cgtcagagat tttggcagcg gcacctacaa gtacatcttc 1380
ggcaccggca ccagactgaa ggtgctggct aatatccaga atccggaccc tgccgtctac 1440
cagctgaggg actcaaagag ctccgataaa agcgtgtgcc tgttcaccga ctttgattct 1500
cagacaaacg tcagccagtc caaggacagt gacgtgtaca ttacagacaa atgcgtgctg 1560
gatatgcgct ccatggactt caagtctaac agtgcagtgg cctggtccaa caagtctgat 1620
ttcgcttgcg caaacgcctt taacaattca atcattccag aggatacctt ctttccaagc 1680
cccgaatcta gttgtgacgt gaagctggtc gagaaatctt tcgaaacaga tactaacctg 1740
aattttcaga atctgagcgt gatcggcttc cgcattctgc tgctgaaagt ggctgggttt 1800
aatctgctga tgactctgcg gctgtggtct tcctaa 1836
<210> 471
<211> 1821
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 54 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 471
atggatactt ggctcgtgtg ctgggccatc ttcagcctgc tgaaagccgg actgaccgag 60
cctgaagtga cacagacacc tagccaccaa gtgacccaga tgggccaaga ggtcatactg 120
agatgcgtgc ccatcagcaa ccacctgtac ttctactggt acagacagat cctgggccag 180
aaggtggaat tcctcgtcag cttctacaac aacgagatca gcgagaagtc cgagatcttc 240
gacgaccagt tcagcgtgga aagacccgac ggcagcaact tcaccctgaa gatcagaagc 300
accaagctcg aggacagcgc catgtacttt tgcgccatca ccgaccggac caactacggc 360
tacacatttg gcagcggcac cagactgaca gtggtcgagg acctgaataa ggtgttccct 420
cctgaagtgg ccgtgtttga gccttctgag gccgagatca gccacacaca gaaagccaca 480
ctcgtgtgcc tggccaccgg cttttttccc gatcacgtgg aactgtcttg gtgggtcaac 540
ggcaaagagg tgcacagcgg cgtctgtacc gatcctcagc ctctgaaaga gcagcccgct 600
ctgaacgaca gcagatactg cctgagcagc agactgagag tgtccgccac cttctggcag 660
aaccccagaa accacttcag atgccaggtg cagttctacg gcctgagcga gaacgatgag 720
tggacccagg atagagccaa gcctgtgaca cagatcgtgt ctgccgaagc ctggggcaga 780
gccgattgtg gctttaccag cgtgtcctat cagcagggcg tgctgtctgc caccatcctg 840
tatgagatcc tgctgggcaa agccactctg tacgccgtgc tggtgtctgc cctggtgctg 900
atggccatgg tcaagagaaa ggactttggc agcggcgcca ccaacttcag cctgcttaaa 960
caggcaggcg acgtggagga gaatcctggc ccaatgaaga agcacctgac cacctttctg 1020
gtcatcctgt ggctgtactt ctacagaggc aacggcaaga accaggtgga acagagccct 1080
cagagcctga tcatcctgga aggcaagaac tgcaccctgc agtgcaacta caccgtgtct 1140
cccttcagca acctgcggtg gtacaagcag gatacaggca gaggccctgt gtctctgacc 1200
atcatgacct tcagcgagaa caccaagagc aacggcagat acaccgccac actggacgcc 1260
gatacaaagc agagcagcct gcacatcaca gccagccagc tgagcgatag cgccagctac 1320
atctgcgtgg tgtccagaga caactacggc cagaacttcg tgttcggccc tggcacaaga 1380
ctgagcgtgc tgccttatat ccagaatccg gaccctgccg tctaccagct gagggactca 1440
aagagctccg ataaaagcgt gtgcctgttc accgactttg attctcagac aaacgtcagc 1500
cagtccaagg acagtgacgt gtacattaca gacaaatgcg tgctggatat gcgctccatg 1560
gacttcaagt ctaacagtgc agtggcctgg tccaacaagt ctgatttcgc ttgcgcaaac 1620
gcctttaaca attcaatcat tccagaggat accttctttc caagccccga atctagttgt 1680
gacgtgaagc tggtcgagaa atctttcgaa acagatacta acctgaattt tcagaatctg 1740
agcgtgatcg gcttccgcat tctgctgctg aaagtggctg ggtttaatct gctgatgact 1800
ctgcggctgt ggtcttccta a 1821
<210> 472
<211> 1821
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 55 Кодон-оптимизированная/цистеин-модифицированная полноразмерная
последовательность
<400> 472
atggatactt ggctcgtgtg ctgggccatc ttcagcctgc tgaaagccgg actgaccgag 60
cctgaagtga cacagacacc tagccaccaa gtgacccaga tgggccaaga ggtcatactg 120
agatgcgtgc ccatcagcaa ccacctgtac ttctactggt acagacagat cctgggccag 180
aaggtggaat tcctcgtcag cttctacaac aacgagatca gcgagaagtc cgagatcttc 240
gacgaccagt tcagcgtgga aagacccgac ggcagcaact tcaccctgaa gatcagaagc 300
accaagctcg aggacagcgc catgtacttt tgcgccagca ccaccagaag cagctacgag 360
cagtatttcg gccctggcac cagactgacc gtgacagaag atctgaagaa cgtcttccct 420
cctgaagtgg ccgtgtttga gccttctgag gccgagatca gccacacaca gaaagccaca 480
ctcgtgtgtc tggccaccgg cttctatccc gatcacgtgg aactgtcttg gtgggtcaac 540
ggcaaagagg tgcacagcgg cgtctgtacc gatcctcagc ctctgaaaga gcagcccgct 600
ctgaacgaca gcagatactg cctgagcagc agactgagag tgtccgccac cttctggcag 660
aaccccagaa accacttcag atgccaggtg cagttctacg gcctgagcga gaacgatgag 720
tggacccagg atagagccaa gcctgtgaca cagatcgtgt ctgccgaagc ctggggcaga 780
gccgattgtg gctttaccag cgagagctac cagcagggcg tgctgtctgc cacaatcctg 840
tacgagatcc tgctgggcaa agccactctg tacgccgtgc tggtgtctgc cctggtgctg 900
atggccatgg tcaagcggaa ggatagcaga ggcggaagcg gcgccacaaa cttctcactg 960
ctgaaacagg ccggcgacgt ggaggagaat cctggcccaa tgaagacctt cgccggcttc 1020
agcttcctgt tcctgtggct gcagctggac tgtatgagca gaggcgagga tgtggaacag 1080
agcctgtttc tgagcgtcag agaaggcgac agcagcgtga tcaactgcac ctacaccgac 1140
agctccagca cctacctgta ctggtacaag caagaacctg gcgccggact gcagctgctg 1200
acctacatct tcagcaacat ggacatgaag caggaccagc ggctgaccgt gctgctgaac 1260
aagaaggata agcacctgag cctgcggatc gccgatacac agacaggcga ctccgccatc 1320
tacttttgtg ctgtgcctag cggcgccacc aacaagctga tctttggaac aggcacactg 1380
ctggccgtgc agcccaatat ccagaatccg gaccctgccg tctaccagct gagggactca 1440
aagagctccg ataaaagcgt gtgcctgttc accgactttg attctcagac aaacgtcagc 1500
cagtccaagg acagtgacgt gtacattaca gacaaatgcg tgctggatat gcgctccatg 1560
gacttcaagt ctaacagtgc agtggcctgg tccaacaagt ctgatttcgc ttgcgcaaac 1620
gcctttaaca attcaatcat tccagaggat accttctttc caagccccga atctagttgt 1680
gacgtgaagc tggtcgagaa atctttcgaa acagatacta acctgaattt tcagaatctg 1740
agcgtgatcg gcttccgcat tctgctgctg aaagtggctg ggtttaatct gctgatgact 1800
ctgcggctgt ggtcttccta a 1821
<210> 473
<211> 255
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 15 - альфа нативный
<400> 473
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
1 5 10 15
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln Ser Tyr
20 25 30
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
35 40 45
Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly Arg Tyr
50 55 60
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
65 70 75 80
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Lys Pro
85 90 95
Gly Gly Tyr Asn Lys Leu Ile Phe Gly Ala Gly Thr Arg Leu Ala Val
100 105 110
His Pro Tyr Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp
115 120 125
Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser
130 135 140
Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp
145 150 155 160
Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala
165 170 175
Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn
180 185 190
Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser
195 200 205
Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu
210 215 220
Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys
225 230 235 240
Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250 255
<210> 474
<211> 255
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 15 - альфа, цистеин-модифицированный
<400> 474
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
1 5 10 15
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln Ser Tyr
20 25 30
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
35 40 45
Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly Arg Tyr
50 55 60
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
65 70 75 80
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Lys Pro
85 90 95
Gly Gly Tyr Asn Lys Leu Ile Phe Gly Ala Gly Thr Arg Leu Ala Val
100 105 110
His Pro Tyr Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp
115 120 125
Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser
130 135 140
Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp
145 150 155 160
Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala
165 170 175
Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn
180 185 190
Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser
195 200 205
Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu
210 215 220
Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys
225 230 235 240
Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250 255
<210> 475
<211> 275
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 15 - альфа нативный
<400> 475
Met Ser Leu Ser Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu
1 5 10 15
Gly Pro Gly Ile Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe
20 25 30
Val Gln Glu Lys Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser
35 40 45
Asp Gln Ser Tyr Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu
50 55 60
Met Ile Phe Leu Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr
65 70 75 80
Glu Gly Arg Tyr Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn
85 90 95
Leu Val Ile Ser Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys
100 105 110
Ala Met Lys Pro Gly Gly Tyr Asn Lys Leu Ile Phe Gly Ala Gly Thr
115 120 125
Arg Leu Ala Val His Pro Tyr Ile Gln Asn Pro Asp Pro Ala Val Tyr
130 135 140
Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr
145 150 155 160
Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val
165 170 175
Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys
180 185 190
Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala
195 200 205
Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser
210 215 220
Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr
225 230 235 240
Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile
245 250 255
Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu
260 265 270
Trp Ser Ser
275
<210> 476
<211> 275
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 15 - альфа, цистеин-модифицированный
<400> 476
Met Ser Leu Ser Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu
1 5 10 15
Gly Pro Gly Ile Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe
20 25 30
Val Gln Glu Lys Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser
35 40 45
Asp Gln Ser Tyr Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu
50 55 60
Met Ile Phe Leu Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr
65 70 75 80
Glu Gly Arg Tyr Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn
85 90 95
Leu Val Ile Ser Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys
100 105 110
Ala Met Lys Pro Gly Gly Tyr Asn Lys Leu Ile Phe Gly Ala Gly Thr
115 120 125
Arg Leu Ala Val His Pro Tyr Ile Gln Asn Pro Asp Pro Ala Val Tyr
130 135 140
Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr
145 150 155 160
Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val
165 170 175
Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys
180 185 190
Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala
195 200 205
Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser
210 215 220
Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr
225 230 235 240
Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile
245 250 255
Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu
260 265 270
Trp Ser Ser
275
<210> 477
<211> 114
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 15 альфа, вариабельная область
<400> 477
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
1 5 10 15
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln Ser Tyr
20 25 30
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
35 40 45
Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly Arg Tyr
50 55 60
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
65 70 75 80
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Lys Pro
85 90 95
Gly Gly Tyr Asn Lys Leu Ile Phe Gly Ala Gly Thr Arg Leu Ala Val
100 105 110
His Pro
<210> 478
<211> 11
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 15 альфа CDR3
<400> 478
Ala Met Lys Pro Gly Gly Tyr Asn Lys Leu Ile
1 5 10
<210> 479
<211> 294
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 15 - бета нативный
<400> 479
Asp Ala Gly Val Ile Gln Ser Pro Arg His Glu Val Thr Glu Met Gly
1 5 10 15
Gln Glu Val Thr Leu Arg Cys Lys Pro Ile Ser Gly His Asn Ser Leu
20 25 30
Phe Trp Tyr Arg Gln Thr Met Met Arg Gly Leu Glu Leu Leu Ile Tyr
35 40 45
Phe Asn Asn Asn Val Pro Ile Asp Asp Ser Gly Met Pro Glu Asp Arg
50 55 60
Phe Ser Ala Lys Met Pro Asn Ala Ser Phe Ser Thr Leu Lys Ile Gln
65 70 75 80
Pro Ser Glu Pro Arg Asp Ser Ala Val Tyr Phe Cys Ala Ser Ser Leu
85 90 95
Val Gly Arg Ser Arg Thr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu
100 105 110
Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val
115 120 125
Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu
130 135 140
Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp
145 150 155 160
Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln
165 170 175
Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser
180 185 190
Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His
195 200 205
Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp
210 215 220
Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala
225 230 235 240
Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly
245 250 255
Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr
260 265 270
Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys
275 280 285
Arg Lys Asp Ser Arg Gly
290
<210> 480
<211> 294
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 15 - бета, цистеин-модифицированный
<400> 480
Asp Ala Gly Val Ile Gln Ser Pro Arg His Glu Val Thr Glu Met Gly
1 5 10 15
Gln Glu Val Thr Leu Arg Cys Lys Pro Ile Ser Gly His Asn Ser Leu
20 25 30
Phe Trp Tyr Arg Gln Thr Met Met Arg Gly Leu Glu Leu Leu Ile Tyr
35 40 45
Phe Asn Asn Asn Val Pro Ile Asp Asp Ser Gly Met Pro Glu Asp Arg
50 55 60
Phe Ser Ala Lys Met Pro Asn Ala Ser Phe Ser Thr Leu Lys Ile Gln
65 70 75 80
Pro Ser Glu Pro Arg Asp Ser Ala Val Tyr Phe Cys Ala Ser Ser Leu
85 90 95
Val Gly Arg Ser Arg Thr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu
100 105 110
Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val
115 120 125
Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu
130 135 140
Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp
145 150 155 160
Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln
165 170 175
Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser
180 185 190
Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His
195 200 205
Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp
210 215 220
Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala
225 230 235 240
Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly
245 250 255
Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr
260 265 270
Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys
275 280 285
Arg Lys Asp Ser Arg Gly
290
<210> 481
<211> 313
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 15 - бета нативный
<400> 481
Met Asp Ser Trp Thr Phe Cys Cys Val Ser Leu Cys Ile Leu Val Ala
1 5 10 15
Lys His Thr Asp Ala Gly Val Ile Gln Ser Pro Arg His Glu Val Thr
20 25 30
Glu Met Gly Gln Glu Val Thr Leu Arg Cys Lys Pro Ile Ser Gly His
35 40 45
Asn Ser Leu Phe Trp Tyr Arg Gln Thr Met Met Arg Gly Leu Glu Leu
50 55 60
Leu Ile Tyr Phe Asn Asn Asn Val Pro Ile Asp Asp Ser Gly Met Pro
65 70 75 80
Glu Asp Arg Phe Ser Ala Lys Met Pro Asn Ala Ser Phe Ser Thr Leu
85 90 95
Lys Ile Gln Pro Ser Glu Pro Arg Asp Ser Ala Val Tyr Phe Cys Ala
100 105 110
Ser Ser Leu Val Gly Arg Ser Arg Thr Glu Gln Tyr Phe Gly Pro Gly
115 120 125
Thr Arg Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu
130 135 140
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
145 150 155 160
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu
165 170 175
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr
180 185 190
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
195 200 205
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
210 215 220
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
225 230 235 240
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
245 250 255
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr
260 265 270
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
275 280 285
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
290 295 300
Met Val Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 482
<211> 313
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 15 - бета, цистеин-модифицированный
<400> 482
Met Asp Ser Trp Thr Phe Cys Cys Val Ser Leu Cys Ile Leu Val Ala
1 5 10 15
Lys His Thr Asp Ala Gly Val Ile Gln Ser Pro Arg His Glu Val Thr
20 25 30
Glu Met Gly Gln Glu Val Thr Leu Arg Cys Lys Pro Ile Ser Gly His
35 40 45
Asn Ser Leu Phe Trp Tyr Arg Gln Thr Met Met Arg Gly Leu Glu Leu
50 55 60
Leu Ile Tyr Phe Asn Asn Asn Val Pro Ile Asp Asp Ser Gly Met Pro
65 70 75 80
Glu Asp Arg Phe Ser Ala Lys Met Pro Asn Ala Ser Phe Ser Thr Leu
85 90 95
Lys Ile Gln Pro Ser Glu Pro Arg Asp Ser Ala Val Tyr Phe Cys Ala
100 105 110
Ser Ser Leu Val Gly Arg Ser Arg Thr Glu Gln Tyr Phe Gly Pro Gly
115 120 125
Thr Arg Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu
130 135 140
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
145 150 155 160
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu
165 170 175
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr
180 185 190
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
195 200 205
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
210 215 220
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
225 230 235 240
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
245 250 255
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr
260 265 270
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
275 280 285
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
290 295 300
Met Val Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 483
<211> 115
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 15 бета, вариабельная область
<400> 483
Asp Ala Gly Val Ile Gln Ser Pro Arg His Glu Val Thr Glu Met Gly
1 5 10 15
Gln Glu Val Thr Leu Arg Cys Lys Pro Ile Ser Gly His Asn Ser Leu
20 25 30
Phe Trp Tyr Arg Gln Thr Met Met Arg Gly Leu Glu Leu Leu Ile Tyr
35 40 45
Phe Asn Asn Asn Val Pro Ile Asp Asp Ser Gly Met Pro Glu Asp Arg
50 55 60
Phe Ser Ala Lys Met Pro Asn Ala Ser Phe Ser Thr Leu Lys Ile Gln
65 70 75 80
Pro Ser Glu Pro Arg Asp Ser Ala Val Tyr Phe Cys Ala Ser Ser Leu
85 90 95
Val Gly Arg Ser Arg Thr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu
100 105 110
Thr Val Thr
115
<210> 484
<211> 5
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 15 бета CDR1
<400> 484
Ser Gly His Asn Ser
1 5
<210> 485
<211> 6
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 15 бета CDR2
<400> 485
Phe Asn Asn Asn Val Pro
1 5
<210> 486
<211> 13
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 15 бета CDR3
<400> 486
Ala Ser Ser Leu Val Gly Arg Ser Arg Thr Glu Gln Tyr
1 5 10
<210> 487
<211> 19
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 15 - бета, Сигнальный пептид
<400> 487
Met Asp Ser Trp Thr Phe Cys Cys Val Ser Leu Cys Ile Leu Val Ala
1 5 10 15
Lys His Thr
<210> 488
<211> 253
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 16 - альфа нативный
<400> 488
Lys Gln Glu Val Thr Gln Ile Pro Ala Ala Leu Ser Val Pro Glu Gly
1 5 10 15
Glu Asn Leu Val Leu Asn Cys Ser Phe Thr Asp Ser Ala Ile Tyr Asn
20 25 30
Leu Gln Trp Phe Arg Gln Asp Pro Gly Lys Gly Leu Thr Ser Leu Leu
35 40 45
Leu Ile Gln Ser Ser Gln Arg Glu Gln Thr Ser Gly Arg Leu Asn Ala
50 55 60
Ser Leu Asp Lys Ser Ser Gly Arg Ser Thr Leu Tyr Ile Ala Ala Ser
65 70 75 80
Gln Pro Gly Asp Ser Ala Thr Tyr Leu Cys Ala Val Arg Pro Ala Asn
85 90 95
Asn Asn Asp Met Arg Phe Gly Ala Gly Thr Arg Leu Thr Val Lys Pro
100 105 110
Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys
115 120 125
Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr
130 135 140
Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr
145 150 155 160
Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala
165 170 175
Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser
180 185 190
Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp
195 200 205
Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe
210 215 220
Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala
225 230 235 240
Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 489
<211> 253
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 16 - альфа, цистеин-модифицированный
<400> 489
Lys Gln Glu Val Thr Gln Ile Pro Ala Ala Leu Ser Val Pro Glu Gly
1 5 10 15
Glu Asn Leu Val Leu Asn Cys Ser Phe Thr Asp Ser Ala Ile Tyr Asn
20 25 30
Leu Gln Trp Phe Arg Gln Asp Pro Gly Lys Gly Leu Thr Ser Leu Leu
35 40 45
Leu Ile Gln Ser Ser Gln Arg Glu Gln Thr Ser Gly Arg Leu Asn Ala
50 55 60
Ser Leu Asp Lys Ser Ser Gly Arg Ser Thr Leu Tyr Ile Ala Ala Ser
65 70 75 80
Gln Pro Gly Asp Ser Ala Thr Tyr Leu Cys Ala Val Arg Pro Ala Asn
85 90 95
Asn Asn Asp Met Arg Phe Gly Ala Gly Thr Arg Leu Thr Val Lys Pro
100 105 110
Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys
115 120 125
Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr
130 135 140
Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys
145 150 155 160
Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala
165 170 175
Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser
180 185 190
Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp
195 200 205
Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe
210 215 220
Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala
225 230 235 240
Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 490
<211> 272
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 16 - альфа нативный
<400> 490
Met Glu Thr Leu Leu Gly Leu Leu Ile Leu Trp Leu Gln Leu Gln Trp
1 5 10 15
Val Ser Ser Lys Gln Glu Val Thr Gln Ile Pro Ala Ala Leu Ser Val
20 25 30
Pro Glu Gly Glu Asn Leu Val Leu Asn Cys Ser Phe Thr Asp Ser Ala
35 40 45
Ile Tyr Asn Leu Gln Trp Phe Arg Gln Asp Pro Gly Lys Gly Leu Thr
50 55 60
Ser Leu Leu Leu Ile Gln Ser Ser Gln Arg Glu Gln Thr Ser Gly Arg
65 70 75 80
Leu Asn Ala Ser Leu Asp Lys Ser Ser Gly Arg Ser Thr Leu Tyr Ile
85 90 95
Ala Ala Ser Gln Pro Gly Asp Ser Ala Thr Tyr Leu Cys Ala Val Arg
100 105 110
Pro Ala Asn Asn Asn Asp Met Arg Phe Gly Ala Gly Thr Arg Leu Thr
115 120 125
Val Lys Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg
130 135 140
Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp
145 150 155 160
Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr
165 170 175
Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser
180 185 190
Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe
195 200 205
Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser
210 215 220
Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn
225 230 235 240
Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu
245 250 255
Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
260 265 270
<210> 491
<211> 272
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 16 - альфа, цистеин-модифицированный
<400> 491
Met Glu Thr Leu Leu Gly Leu Leu Ile Leu Trp Leu Gln Leu Gln Trp
1 5 10 15
Val Ser Ser Lys Gln Glu Val Thr Gln Ile Pro Ala Ala Leu Ser Val
20 25 30
Pro Glu Gly Glu Asn Leu Val Leu Asn Cys Ser Phe Thr Asp Ser Ala
35 40 45
Ile Tyr Asn Leu Gln Trp Phe Arg Gln Asp Pro Gly Lys Gly Leu Thr
50 55 60
Ser Leu Leu Leu Ile Gln Ser Ser Gln Arg Glu Gln Thr Ser Gly Arg
65 70 75 80
Leu Asn Ala Ser Leu Asp Lys Ser Ser Gly Arg Ser Thr Leu Tyr Ile
85 90 95
Ala Ala Ser Gln Pro Gly Asp Ser Ala Thr Tyr Leu Cys Ala Val Arg
100 105 110
Pro Ala Asn Asn Asn Asp Met Arg Phe Gly Ala Gly Thr Arg Leu Thr
115 120 125
Val Lys Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg
130 135 140
Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp
145 150 155 160
Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr
165 170 175
Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser
180 185 190
Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe
195 200 205
Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser
210 215 220
Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn
225 230 235 240
Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu
245 250 255
Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
260 265 270
<210> 492
<211> 112
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 16 альфа, вариабельная область
<400> 492
Lys Gln Glu Val Thr Gln Ile Pro Ala Ala Leu Ser Val Pro Glu Gly
1 5 10 15
Glu Asn Leu Val Leu Asn Cys Ser Phe Thr Asp Ser Ala Ile Tyr Asn
20 25 30
Leu Gln Trp Phe Arg Gln Asp Pro Gly Lys Gly Leu Thr Ser Leu Leu
35 40 45
Leu Ile Gln Ser Ser Gln Arg Glu Gln Thr Ser Gly Arg Leu Asn Ala
50 55 60
Ser Leu Asp Lys Ser Ser Gly Arg Ser Thr Leu Tyr Ile Ala Ala Ser
65 70 75 80
Gln Pro Gly Asp Ser Ala Thr Tyr Leu Cys Ala Val Arg Pro Ala Asn
85 90 95
Asn Asn Asp Met Arg Phe Gly Ala Gly Thr Arg Leu Thr Val Lys Pro
100 105 110
<210> 493
<211> 11
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 16 альфа CDR3
<400> 493
Ala Val Arg Pro Ala Asn Asn Asn Asp Met Arg
1 5 10
<210> 494
<211> 291
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 16 - бета нативный
<400> 494
Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys Arg Thr Gly
1 5 10 15
Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His Glu Asn Met
20 25 30
Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu Ile Tyr Phe
35 40 45
Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro Glu Gly Tyr
50 55 60
Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile Leu Glu Ser
65 70 75 80
Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser Ser Leu Trp
85 90 95
Gly Arg Ser Asn Gln Pro Gln His Phe Gly Asp Gly Thr Arg Leu Ser
100 105 110
Ile Leu Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val Phe
115 120 125
Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val
130 135 140
Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp Trp
145 150 155 160
Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro
165 170 175
Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser
180 185 190
Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe
195 200 205
Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr
210 215 220
Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp
225 230 235 240
Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly Val
245 250 255
Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu
260 265 270
Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg
275 280 285
Lys Asp Phe
290
<210> 495
<211> 291
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 16 / TCR 17 - бета, цистеин-модифицированный
<400> 495
Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys Arg Thr Gly
1 5 10 15
Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His Glu Asn Met
20 25 30
Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu Ile Tyr Phe
35 40 45
Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro Glu Gly Tyr
50 55 60
Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile Leu Glu Ser
65 70 75 80
Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser Ser Leu Trp
85 90 95
Gly Arg Ser Asn Gln Pro Gln His Phe Gly Asp Gly Thr Arg Leu Ser
100 105 110
Ile Leu Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val Phe
115 120 125
Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val
130 135 140
Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp Trp
145 150 155 160
Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro
165 170 175
Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser
180 185 190
Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe
195 200 205
Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr
210 215 220
Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp
225 230 235 240
Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly Val
245 250 255
Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu
260 265 270
Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg
275 280 285
Lys Asp Phe
290
<210> 496
<211> 310
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 16 / TCR 17 - бета нативный
<400> 496
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Ser Leu Trp Gly Arg Ser Asn Gln Pro Gln His Phe Gly Asp Gly Thr
115 120 125
Arg Leu Ser Ile Leu Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe
305 310
<210> 497
<211> 310
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 16 / TCR 17 - бета, цистеин-модифицированный
<400> 497
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Ser Leu Trp Gly Arg Ser Asn Gln Pro Gln His Phe Gly Asp Gly Thr
115 120 125
Arg Leu Ser Ile Leu Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe
305 310
<210> 498
<211> 114
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 16 / TCR 17 - бета, вариабельная область
<400> 498
Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys Arg Thr Gly
1 5 10 15
Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His Glu Asn Met
20 25 30
Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu Ile Tyr Phe
35 40 45
Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro Glu Gly Tyr
50 55 60
Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile Leu Glu Ser
65 70 75 80
Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser Ser Leu Trp
85 90 95
Gly Arg Ser Asn Gln Pro Gln His Phe Gly Asp Gly Thr Arg Leu Ser
100 105 110
Ile Leu
<210> 499
<211> 13
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 16 / TCR 17 бета CDR3
<400> 499
Ala Ser Ser Leu Trp Gly Arg Ser Asn Gln Pro Gln His
1 5 10
<210> 500
<211> 255
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 17 - альфа нативный
<400> 500
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
1 5 10 15
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Pro Ser Tyr
20 25 30
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
35 40 45
Ile Tyr Gln Gly Ser Tyr Asp Gln Gln Asn Ala Thr Glu Gly Arg Tyr
50 55 60
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
65 70 75 80
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Arg Glu
85 90 95
Gly Arg Gly Asp Lys Ile Ile Phe Gly Lys Gly Thr Arg Leu His Ile
100 105 110
Leu Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp
115 120 125
Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser
130 135 140
Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp
145 150 155 160
Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala
165 170 175
Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn
180 185 190
Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser
195 200 205
Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu
210 215 220
Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys
225 230 235 240
Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250 255
<210> 501
<211> 255
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 17 - альфа, цистеин-модифицированный
<400> 501
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
1 5 10 15
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Pro Ser Tyr
20 25 30
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
35 40 45
Ile Tyr Gln Gly Ser Tyr Asp Gln Gln Asn Ala Thr Glu Gly Arg Tyr
50 55 60
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
65 70 75 80
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Arg Glu
85 90 95
Gly Arg Gly Asp Lys Ile Ile Phe Gly Lys Gly Thr Arg Leu His Ile
100 105 110
Leu Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp
115 120 125
Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser
130 135 140
Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp
145 150 155 160
Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala
165 170 175
Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn
180 185 190
Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser
195 200 205
Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu
210 215 220
Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys
225 230 235 240
Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250 255
<210> 502
<211> 275
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 17 - альфа нативный
<400> 502
Met Ser Leu Ser Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu
1 5 10 15
Gly Pro Gly Ile Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe
20 25 30
Val Gln Glu Lys Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser
35 40 45
Asp Pro Ser Tyr Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu
50 55 60
Met Ile Phe Leu Ile Tyr Gln Gly Ser Tyr Asp Gln Gln Asn Ala Thr
65 70 75 80
Glu Gly Arg Tyr Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn
85 90 95
Leu Val Ile Ser Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys
100 105 110
Ala Met Arg Glu Gly Arg Gly Asp Lys Ile Ile Phe Gly Lys Gly Thr
115 120 125
Arg Leu His Ile Leu Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr
130 135 140
Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr
145 150 155 160
Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val
165 170 175
Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys
180 185 190
Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala
195 200 205
Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser
210 215 220
Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr
225 230 235 240
Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile
245 250 255
Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu
260 265 270
Trp Ser Ser
275
<210> 503
<211> 275
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 17 - альфа, цистеин-модифицированный
<400> 503
Met Ser Leu Ser Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu
1 5 10 15
Gly Pro Gly Ile Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe
20 25 30
Val Gln Glu Lys Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser
35 40 45
Asp Pro Ser Tyr Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu
50 55 60
Met Ile Phe Leu Ile Tyr Gln Gly Ser Tyr Asp Gln Gln Asn Ala Thr
65 70 75 80
Glu Gly Arg Tyr Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn
85 90 95
Leu Val Ile Ser Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys
100 105 110
Ala Met Arg Glu Gly Arg Gly Asp Lys Ile Ile Phe Gly Lys Gly Thr
115 120 125
Arg Leu His Ile Leu Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr
130 135 140
Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr
145 150 155 160
Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val
165 170 175
Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys
180 185 190
Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala
195 200 205
Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser
210 215 220
Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr
225 230 235 240
Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile
245 250 255
Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu
260 265 270
Trp Ser Ser
275
<210> 504
<211> 114
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 17 - альфа, вариабельная область
<400> 504
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
1 5 10 15
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Pro Ser Tyr
20 25 30
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
35 40 45
Ile Tyr Gln Gly Ser Tyr Asp Gln Gln Asn Ala Thr Glu Gly Arg Tyr
50 55 60
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
65 70 75 80
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Arg Glu
85 90 95
Gly Arg Gly Asp Lys Ile Ile Phe Gly Lys Gly Thr Arg Leu His Ile
100 105 110
Leu Pro
<210> 505
<211> 11
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 17 альфа CDR3
<400> 505
Ala Met Arg Glu Gly Arg Gly Asp Lys Ile Ile
1 5 10
<210> 506
<211> 256
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 18 - альфа нативный
<400> 506
Lys Gln Glu Val Thr Gln Ile Pro Ala Ala Leu Ser Val Pro Glu Gly
1 5 10 15
Glu Asn Leu Val Leu Asn Cys Ser Phe Thr Asp Ser Ala Ile Tyr Asn
20 25 30
Leu Gln Trp Phe Arg Gln Asp Pro Gly Lys Gly Leu Thr Ser Leu Leu
35 40 45
Leu Ile Gln Ser Ser Gln Arg Glu Gln Thr Ser Gly Arg Leu Asn Ala
50 55 60
Ser Leu Asp Lys Ser Ser Gly Arg Ser Thr Leu Tyr Ile Ala Ala Ser
65 70 75 80
Gln Pro Gly Asp Ser Ala Thr Tyr Leu Cys Ala Glu Gly Asn Ala Gly
85 90 95
Gly Thr Ser Tyr Gly Lys Leu Thr Phe Gly Gln Gly Thr Ile Leu Thr
100 105 110
Val His Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg
115 120 125
Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp
130 135 140
Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr
145 150 155 160
Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser
165 170 175
Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe
180 185 190
Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser
195 200 205
Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn
210 215 220
Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu
225 230 235 240
Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250 255
<210> 507
<211> 256
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 18 - альфа, цистеин-модифицированный
<400> 507
Lys Gln Glu Val Thr Gln Ile Pro Ala Ala Leu Ser Val Pro Glu Gly
1 5 10 15
Glu Asn Leu Val Leu Asn Cys Ser Phe Thr Asp Ser Ala Ile Tyr Asn
20 25 30
Leu Gln Trp Phe Arg Gln Asp Pro Gly Lys Gly Leu Thr Ser Leu Leu
35 40 45
Leu Ile Gln Ser Ser Gln Arg Glu Gln Thr Ser Gly Arg Leu Asn Ala
50 55 60
Ser Leu Asp Lys Ser Ser Gly Arg Ser Thr Leu Tyr Ile Ala Ala Ser
65 70 75 80
Gln Pro Gly Asp Ser Ala Thr Tyr Leu Cys Ala Glu Gly Asn Ala Gly
85 90 95
Gly Thr Ser Tyr Gly Lys Leu Thr Phe Gly Gln Gly Thr Ile Leu Thr
100 105 110
Val His Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg
115 120 125
Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp
130 135 140
Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr
145 150 155 160
Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser
165 170 175
Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe
180 185 190
Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser
195 200 205
Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn
210 215 220
Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu
225 230 235 240
Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250 255
<210> 508
<211> 275
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 18 - альфа нативный
<400> 508
Met Glu Thr Leu Leu Gly Leu Leu Ile Leu Trp Leu Gln Leu Gln Trp
1 5 10 15
Val Ser Ser Lys Gln Glu Val Thr Gln Ile Pro Ala Ala Leu Ser Val
20 25 30
Pro Glu Gly Glu Asn Leu Val Leu Asn Cys Ser Phe Thr Asp Ser Ala
35 40 45
Ile Tyr Asn Leu Gln Trp Phe Arg Gln Asp Pro Gly Lys Gly Leu Thr
50 55 60
Ser Leu Leu Leu Ile Gln Ser Ser Gln Arg Glu Gln Thr Ser Gly Arg
65 70 75 80
Leu Asn Ala Ser Leu Asp Lys Ser Ser Gly Arg Ser Thr Leu Tyr Ile
85 90 95
Ala Ala Ser Gln Pro Gly Asp Ser Ala Thr Tyr Leu Cys Ala Glu Gly
100 105 110
Asn Ala Gly Gly Thr Ser Tyr Gly Lys Leu Thr Phe Gly Gln Gly Thr
115 120 125
Ile Leu Thr Val His Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr
130 135 140
Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr
145 150 155 160
Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val
165 170 175
Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys
180 185 190
Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala
195 200 205
Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser
210 215 220
Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr
225 230 235 240
Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile
245 250 255
Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu
260 265 270
Trp Ser Ser
275
<210> 509
<211> 275
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 18 - альфа, цистеин-модифицированный
<400> 509
Met Glu Thr Leu Leu Gly Leu Leu Ile Leu Trp Leu Gln Leu Gln Trp
1 5 10 15
Val Ser Ser Lys Gln Glu Val Thr Gln Ile Pro Ala Ala Leu Ser Val
20 25 30
Pro Glu Gly Glu Asn Leu Val Leu Asn Cys Ser Phe Thr Asp Ser Ala
35 40 45
Ile Tyr Asn Leu Gln Trp Phe Arg Gln Asp Pro Gly Lys Gly Leu Thr
50 55 60
Ser Leu Leu Leu Ile Gln Ser Ser Gln Arg Glu Gln Thr Ser Gly Arg
65 70 75 80
Leu Asn Ala Ser Leu Asp Lys Ser Ser Gly Arg Ser Thr Leu Tyr Ile
85 90 95
Ala Ala Ser Gln Pro Gly Asp Ser Ala Thr Tyr Leu Cys Ala Glu Gly
100 105 110
Asn Ala Gly Gly Thr Ser Tyr Gly Lys Leu Thr Phe Gly Gln Gly Thr
115 120 125
Ile Leu Thr Val His Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr
130 135 140
Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr
145 150 155 160
Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val
165 170 175
Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys
180 185 190
Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala
195 200 205
Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser
210 215 220
Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr
225 230 235 240
Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile
245 250 255
Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu
260 265 270
Trp Ser Ser
275
<210> 510
<211> 115
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 18 альфа, вариабельная область
<400> 510
Lys Gln Glu Val Thr Gln Ile Pro Ala Ala Leu Ser Val Pro Glu Gly
1 5 10 15
Glu Asn Leu Val Leu Asn Cys Ser Phe Thr Asp Ser Ala Ile Tyr Asn
20 25 30
Leu Gln Trp Phe Arg Gln Asp Pro Gly Lys Gly Leu Thr Ser Leu Leu
35 40 45
Leu Ile Gln Ser Ser Gln Arg Glu Gln Thr Ser Gly Arg Leu Asn Ala
50 55 60
Ser Leu Asp Lys Ser Ser Gly Arg Ser Thr Leu Tyr Ile Ala Ala Ser
65 70 75 80
Gln Pro Gly Asp Ser Ala Thr Tyr Leu Cys Ala Glu Gly Asn Ala Gly
85 90 95
Gly Thr Ser Tyr Gly Lys Leu Thr Phe Gly Gln Gly Thr Ile Leu Thr
100 105 110
Val His Pro
115
<210> 511
<211> 14
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 18 альфа CDR3
<400> 511
Ala Glu Gly Asn Ala Gly Gly Thr Ser Tyr Gly Lys Leu Thr
1 5 10
<210> 512
<211> 291
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 18 - бета нативный
<400> 512
Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys Arg Thr Gly
1 5 10 15
Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His Glu Asn Met
20 25 30
Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu Ile Tyr Phe
35 40 45
Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro Glu Gly Tyr
50 55 60
Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile Leu Glu Ser
65 70 75 80
Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser Ser Phe Trp
85 90 95
Gly Arg Ser Asn Ser Pro Leu His Phe Gly Asn Gly Thr Arg Leu Thr
100 105 110
Val Thr Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val Phe
115 120 125
Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val
130 135 140
Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp Trp
145 150 155 160
Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro
165 170 175
Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser
180 185 190
Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe
195 200 205
Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr
210 215 220
Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp
225 230 235 240
Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly Val
245 250 255
Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu
260 265 270
Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg
275 280 285
Lys Asp Phe
290
<210> 513
<211> 291
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 18 - бета, цистеин-модифицированный
<400> 513
Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys Arg Thr Gly
1 5 10 15
Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His Glu Asn Met
20 25 30
Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu Ile Tyr Phe
35 40 45
Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro Glu Gly Tyr
50 55 60
Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile Leu Glu Ser
65 70 75 80
Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser Ser Phe Trp
85 90 95
Gly Arg Ser Asn Ser Pro Leu His Phe Gly Asn Gly Thr Arg Leu Thr
100 105 110
Val Thr Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val Phe
115 120 125
Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val
130 135 140
Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp Trp
145 150 155 160
Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro
165 170 175
Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser
180 185 190
Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe
195 200 205
Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr
210 215 220
Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp
225 230 235 240
Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly Val
245 250 255
Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu
260 265 270
Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg
275 280 285
Lys Asp Phe
290
<210> 514
<211> 310
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 18 - бета нативный
<400> 514
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Ser Phe Trp Gly Arg Ser Asn Ser Pro Leu His Phe Gly Asn Gly Thr
115 120 125
Arg Leu Thr Val Thr Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe
305 310
<210> 515
<211> 310
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 18 - бета, цистеин-модифицированный
<400> 515
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Ser Phe Trp Gly Arg Ser Asn Ser Pro Leu His Phe Gly Asn Gly Thr
115 120 125
Arg Leu Thr Val Thr Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe
305 310
<210> 516
<211> 114
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 18 бета, вариабельная область
<400> 516
Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys Arg Thr Gly
1 5 10 15
Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His Glu Asn Met
20 25 30
Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu Ile Tyr Phe
35 40 45
Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro Glu Gly Tyr
50 55 60
Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile Leu Glu Ser
65 70 75 80
Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser Ser Phe Trp
85 90 95
Gly Arg Ser Asn Ser Pro Leu His Phe Gly Asn Gly Thr Arg Leu Thr
100 105 110
Val Thr
<210> 517
<211> 13
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 18 бета CDR3
<400> 517
Ala Ser Ser Phe Trp Gly Arg Ser Asn Ser Pro Leu His
1 5 10
<210> 518
<211> 257
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 19 - альфа нативный
<400> 518
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
1 5 10 15
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln Ser Tyr
20 25 30
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
35 40 45
Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly Arg Tyr
50 55 60
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
65 70 75 80
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Arg Glu
85 90 95
Asn Thr Gly Thr Ala Ser Lys Leu Thr Phe Gly Thr Gly Thr Arg Leu
100 105 110
Gln Val Thr Leu Asp Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu
115 120 125
Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe
130 135 140
Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile
145 150 155 160
Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn
165 170 175
Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala
180 185 190
Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu
195 200 205
Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr
210 215 220
Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu
225 230 235 240
Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser
245 250 255
Ser
<210> 519
<211> 257
<212> Белок
<213> Homoe sapiens
<220>
<223> TCR 19 - альфа, цистеин-модифицированный
<400> 519
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
1 5 10 15
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln Ser Tyr
20 25 30
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
35 40 45
Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly Arg Tyr
50 55 60
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
65 70 75 80
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Arg Glu
85 90 95
Asn Thr Gly Thr Ala Ser Lys Leu Thr Phe Gly Thr Gly Thr Arg Leu
100 105 110
Gln Val Thr Leu Asp Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu
115 120 125
Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe
130 135 140
Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile
145 150 155 160
Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn
165 170 175
Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala
180 185 190
Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu
195 200 205
Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr
210 215 220
Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu
225 230 235 240
Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser
245 250 255
Ser
<210> 520
<211> 277
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 19 - альфа нативный
<400> 520
Met Ser Leu Ser Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu
1 5 10 15
Gly Pro Gly Ile Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe
20 25 30
Val Gln Glu Lys Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser
35 40 45
Asp Gln Ser Tyr Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu
50 55 60
Met Ile Phe Leu Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr
65 70 75 80
Glu Gly Arg Tyr Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn
85 90 95
Leu Val Ile Ser Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys
100 105 110
Ala Met Arg Glu Asn Thr Gly Thr Ala Ser Lys Leu Thr Phe Gly Thr
115 120 125
Gly Thr Arg Leu Gln Val Thr Leu Asp Ile Gln Asn Pro Asp Pro Ala
130 135 140
Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu
145 150 155 160
Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser
165 170 175
Asp Val Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp
180 185 190
Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala
195 200 205
Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe
210 215 220
Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe
225 230 235 240
Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe
245 250 255
Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu
260 265 270
Arg Leu Trp Ser Ser
275
<210> 521
<211> 277
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 19 - альфа, цистеин-модифицированный
<400> 521
Met Ser Leu Ser Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu
1 5 10 15
Gly Pro Gly Ile Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe
20 25 30
Val Gln Glu Lys Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser
35 40 45
Asp Gln Ser Tyr Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu
50 55 60
Met Ile Phe Leu Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr
65 70 75 80
Glu Gly Arg Tyr Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn
85 90 95
Leu Val Ile Ser Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys
100 105 110
Ala Met Arg Glu Asn Thr Gly Thr Ala Ser Lys Leu Thr Phe Gly Thr
115 120 125
Gly Thr Arg Leu Gln Val Thr Leu Asp Ile Gln Asn Pro Asp Pro Ala
130 135 140
Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu
145 150 155 160
Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser
165 170 175
Asp Val Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp
180 185 190
Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala
195 200 205
Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe
210 215 220
Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe
225 230 235 240
Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe
245 250 255
Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu
260 265 270
Arg Leu Trp Ser Ser
275
<210> 522
<211> 116
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 19 альфа, вариабельная область
<400> 522
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
1 5 10 15
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln Ser Tyr
20 25 30
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
35 40 45
Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly Arg Tyr
50 55 60
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
65 70 75 80
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Arg Glu
85 90 95
Asn Thr Gly Thr Ala Ser Lys Leu Thr Phe Gly Thr Gly Thr Arg Leu
100 105 110
Gln Val Thr Leu
115
<210> 523
<211> 13
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 19 альфа CDR3
<400> 523
Ala Met Arg Glu Asn Thr Gly Thr Ala Ser Lys Leu Thr
1 5 10
<210> 524
<211> 141
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 19/ TCR 22/ TCR 23/ TCR 24/ TCR 25/ TCR 47
нативный TCR альфа, Константная область
<400> 524
Asp Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys
1 5 10 15
Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr
20 25 30
Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr
35 40 45
Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala
50 55 60
Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser
65 70 75 80
Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp
85 90 95
Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe
100 105 110
Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala
115 120 125
Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
130 135 140
<210> 525
<211> 141
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 19/ TCR 22/ TCR 23/ TCR 24/ TCR 25/ TCR 29/
TCR 47 альфа, Константная область
<400> 525
Asp Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys
1 5 10 15
Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr
20 25 30
Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys
35 40 45
Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala
50 55 60
Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser
65 70 75 80
Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp
85 90 95
Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe
100 105 110
Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala
115 120 125
Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
130 135 140
<210> 526
<211> 293
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 19 - бета нативный
<400> 526
Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys Arg Thr Gly
1 5 10 15
Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His Glu Asn Met
20 25 30
Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu Ile Tyr Phe
35 40 45
Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro Glu Gly Tyr
50 55 60
Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile Leu Glu Ser
65 70 75 80
Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser Ser Ser Trp
85 90 95
Gly Gln Ser Thr Gly Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr
100 105 110
Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe
115 120 125
Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val
130 135 140
Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp
145 150 155 160
Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro
165 170 175
Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser
180 185 190
Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe
195 200 205
Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr
210 215 220
Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp
225 230 235 240
Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val
245 250 255
Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu
260 265 270
Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg
275 280 285
Lys Asp Ser Arg Gly
290
<210> 527
<211> 293
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 19 - бета, цистеин-модифицированный
<400> 527
Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys Arg Thr Gly
1 5 10 15
Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His Glu Asn Met
20 25 30
Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu Ile Tyr Phe
35 40 45
Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro Glu Gly Tyr
50 55 60
Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile Leu Glu Ser
65 70 75 80
Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser Ser Ser Trp
85 90 95
Gly Gln Ser Thr Gly Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr
100 105 110
Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe
115 120 125
Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val
130 135 140
Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp
145 150 155 160
Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro
165 170 175
Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser
180 185 190
Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe
195 200 205
Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr
210 215 220
Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp
225 230 235 240
Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val
245 250 255
Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu
260 265 270
Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg
275 280 285
Lys Asp Ser Arg Gly
290
<210> 528
<211> 312
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 19 - бета нативный
<400> 528
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Ser Ser Trp Gly Gln Ser Thr Gly Glu Gln Tyr Phe Gly Pro Gly Thr
115 120 125
Arg Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 529
<211> 312
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 19 - бета, цистеин-модифицированный
<400> 529
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Ser Ser Trp Gly Gln Ser Thr Gly Glu Gln Tyr Phe Gly Pro Gly Thr
115 120 125
Arg Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 530
<211> 114
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 19 бета, вариабельная область
<400> 530
Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys Arg Thr Gly
1 5 10 15
Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His Glu Asn Met
20 25 30
Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu Ile Tyr Phe
35 40 45
Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro Glu Gly Tyr
50 55 60
Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile Leu Glu Ser
65 70 75 80
Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser Ser Ser Trp
85 90 95
Gly Gln Ser Thr Gly Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr
100 105 110
Val Thr
<210> 531
<211> 13
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 19 бета CDR3
<400> 531
Ala Ser Ser Ser Trp Gly Gln Ser Thr Gly Glu Gln Tyr
1 5 10
<210> 532
<211> 254
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 20 - альфа нативный
<400> 532
Ala Gln Lys Val Thr Gln Ala Gln Thr Glu Ile Ser Val Val Glu Lys
1 5 10 15
Glu Asp Val Thr Leu Asp Cys Val Tyr Glu Thr Arg Asp Thr Thr Tyr
20 25 30
Tyr Leu Phe Trp Tyr Lys Gln Pro Pro Ser Gly Glu Leu Val Phe Leu
35 40 45
Ile Arg Arg Asn Ser Phe Asp Glu Gln Asn Glu Ile Ser Gly Arg Tyr
50 55 60
Ser Trp Asn Phe Gln Lys Ser Thr Ser Ser Phe Asn Phe Thr Ile Thr
65 70 75 80
Ala Ser Gln Val Val Asp Ser Ala Val Tyr Phe Cys Ala Leu Trp Thr
85 90 95
Gly Ala Asn Asn Leu Phe Phe Gly Thr Gly Thr Arg Leu Thr Val Ile
100 105 110
Pro Tyr Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser
115 120 125
Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln
130 135 140
Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys
145 150 155 160
Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val
165 170 175
Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn
180 185 190
Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys
195 200 205
Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn
210 215 220
Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val
225 230 235 240
Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 533
<211> 254
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 20 - альфа, цистеин-модифицированный
<400> 533
Ala Gln Lys Val Thr Gln Ala Gln Thr Glu Ile Ser Val Val Glu Lys
1 5 10 15
Glu Asp Val Thr Leu Asp Cys Val Tyr Glu Thr Arg Asp Thr Thr Tyr
20 25 30
Tyr Leu Phe Trp Tyr Lys Gln Pro Pro Ser Gly Glu Leu Val Phe Leu
35 40 45
Ile Arg Arg Asn Ser Phe Asp Glu Gln Asn Glu Ile Ser Gly Arg Tyr
50 55 60
Ser Trp Asn Phe Gln Lys Ser Thr Ser Ser Phe Asn Phe Thr Ile Thr
65 70 75 80
Ala Ser Gln Val Val Asp Ser Ala Val Tyr Phe Cys Ala Leu Trp Thr
85 90 95
Gly Ala Asn Asn Leu Phe Phe Gly Thr Gly Thr Arg Leu Thr Val Ile
100 105 110
Pro Tyr Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser
115 120 125
Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln
130 135 140
Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys
145 150 155 160
Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val
165 170 175
Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn
180 185 190
Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys
195 200 205
Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn
210 215 220
Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val
225 230 235 240
Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 534
<211> 276
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 20 - альфа нативный
<400> 534
Met Asn Met Leu Thr Ala Ser Leu Leu Arg Ala Val Ile Ala Ser Ile
1 5 10 15
Cys Val Val Ser Ser Met Ala Gln Lys Val Thr Gln Ala Gln Thr Glu
20 25 30
Ile Ser Val Val Glu Lys Glu Asp Val Thr Leu Asp Cys Val Tyr Glu
35 40 45
Thr Arg Asp Thr Thr Tyr Tyr Leu Phe Trp Tyr Lys Gln Pro Pro Ser
50 55 60
Gly Glu Leu Val Phe Leu Ile Arg Arg Asn Ser Phe Asp Glu Gln Asn
65 70 75 80
Glu Ile Ser Gly Arg Tyr Ser Trp Asn Phe Gln Lys Ser Thr Ser Ser
85 90 95
Phe Asn Phe Thr Ile Thr Ala Ser Gln Val Val Asp Ser Ala Val Tyr
100 105 110
Phe Cys Ala Leu Trp Thr Gly Ala Asn Asn Leu Phe Phe Gly Thr Gly
115 120 125
Thr Arg Leu Thr Val Ile Pro Tyr Ile Gln Asn Pro Asp Pro Ala Val
130 135 140
Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe
145 150 155 160
Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp
165 170 175
Val Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe
180 185 190
Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys
195 200 205
Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro
210 215 220
Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu
225 230 235 240
Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg
245 250 255
Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg
260 265 270
Leu Trp Ser Ser
275
<210> 535
<211> 276
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 20 - альфа, цистеин-модифицированный
<400> 535
Met Asn Met Leu Thr Ala Ser Leu Leu Arg Ala Val Ile Ala Ser Ile
1 5 10 15
Cys Val Val Ser Ser Met Ala Gln Lys Val Thr Gln Ala Gln Thr Glu
20 25 30
Ile Ser Val Val Glu Lys Glu Asp Val Thr Leu Asp Cys Val Tyr Glu
35 40 45
Thr Arg Asp Thr Thr Tyr Tyr Leu Phe Trp Tyr Lys Gln Pro Pro Ser
50 55 60
Gly Glu Leu Val Phe Leu Ile Arg Arg Asn Ser Phe Asp Glu Gln Asn
65 70 75 80
Glu Ile Ser Gly Arg Tyr Ser Trp Asn Phe Gln Lys Ser Thr Ser Ser
85 90 95
Phe Asn Phe Thr Ile Thr Ala Ser Gln Val Val Asp Ser Ala Val Tyr
100 105 110
Phe Cys Ala Leu Trp Thr Gly Ala Asn Asn Leu Phe Phe Gly Thr Gly
115 120 125
Thr Arg Leu Thr Val Ile Pro Tyr Ile Gln Asn Pro Asp Pro Ala Val
130 135 140
Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe
145 150 155 160
Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp
165 170 175
Val Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe
180 185 190
Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys
195 200 205
Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro
210 215 220
Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu
225 230 235 240
Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg
245 250 255
Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg
260 265 270
Leu Trp Ser Ser
275
<210> 536
<211> 113
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 20
альфа, вариабельная область
<400> 536
Ala Gln Lys Val Thr Gln Ala Gln Thr Glu Ile Ser Val Val Glu Lys
1 5 10 15
Glu Asp Val Thr Leu Asp Cys Val Tyr Glu Thr Arg Asp Thr Thr Tyr
20 25 30
Tyr Leu Phe Trp Tyr Lys Gln Pro Pro Ser Gly Glu Leu Val Phe Leu
35 40 45
Ile Arg Arg Asn Ser Phe Asp Glu Gln Asn Glu Ile Ser Gly Arg Tyr
50 55 60
Ser Trp Asn Phe Gln Lys Ser Thr Ser Ser Phe Asn Phe Thr Ile Thr
65 70 75 80
Ala Ser Gln Val Val Asp Ser Ala Val Tyr Phe Cys Ala Leu Trp Thr
85 90 95
Gly Ala Asn Asn Leu Phe Phe Gly Thr Gly Thr Arg Leu Thr Val Ile
100 105 110
Pro
<210> 537
<211> 7
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 20 альфа CDR1
<400> 537
Thr Arg Asp Thr Thr Tyr Tyr
1 5
<210> 538
<211> 8
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 20 альфа CDR2
<400> 538
Arg Asn Ser Phe Asp Glu Gln Asn
1 5
<210> 539
<211> 10
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 20 альфа CDR3
<400> 539
Ala Leu Trp Thr Gly Ala Asn Asn Leu Phe
1 5 10
<210> 540
<211> 22
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 20 альфа, Сигнальный пептид
<400> 540
Met Asn Met Leu Thr Ala Ser Leu Leu Arg Ala Val Ile Ala Ser Ile
1 5 10 15
Cys Val Val Ser Ser Met
20
<210> 541
<211> 296
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 20 - бета нативный
<400> 541
Gly Ala Val Val Ser Gln His Pro Ser Arg Val Ile Cys Lys Ser Gly
1 5 10 15
Thr Ser Val Lys Ile Glu Cys Arg Ser Leu Asp Phe Gln Ala Thr Thr
20 25 30
Met Phe Trp Tyr Arg Gln Phe Pro Lys Gln Ser Leu Met Leu Met Ala
35 40 45
Thr Ser Asn Glu Gly Ser Lys Ala Thr Tyr Glu Gln Gly Val Glu Lys
50 55 60
Asp Lys Phe Leu Ile Asn His Ala Ser Leu Thr Leu Ser Thr Leu Thr
65 70 75 80
Val Thr Ser Ala His Pro Glu Asp Ser Ser Phe Tyr Ile Cys Ser Ala
85 90 95
Ser Ser Leu Ala Arg Arg Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr
100 105 110
Arg Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val
115 120 125
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
130 135 140
Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu
145 150 155 160
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp
165 170 175
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
180 185 190
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
195 200 205
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
210 215 220
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
225 230 235 240
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln
245 250 255
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
260 265 270
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
275 280 285
Val Lys Arg Lys Asp Ser Arg Gly
290 295
<210> 542
<211> 296
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 20 - бета, цистеин-модифицированный
<400> 542
Gly Ala Val Val Ser Gln His Pro Ser Arg Val Ile Cys Lys Ser Gly
1 5 10 15
Thr Ser Val Lys Ile Glu Cys Arg Ser Leu Asp Phe Gln Ala Thr Thr
20 25 30
Met Phe Trp Tyr Arg Gln Phe Pro Lys Gln Ser Leu Met Leu Met Ala
35 40 45
Thr Ser Asn Glu Gly Ser Lys Ala Thr Tyr Glu Gln Gly Val Glu Lys
50 55 60
Asp Lys Phe Leu Ile Asn His Ala Ser Leu Thr Leu Ser Thr Leu Thr
65 70 75 80
Val Thr Ser Ala His Pro Glu Asp Ser Ser Phe Tyr Ile Cys Ser Ala
85 90 95
Ser Ser Leu Ala Arg Arg Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr
100 105 110
Arg Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val
115 120 125
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
130 135 140
Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu
145 150 155 160
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp
165 170 175
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
180 185 190
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
195 200 205
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
210 215 220
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
225 230 235 240
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln
245 250 255
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
260 265 270
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
275 280 285
Val Lys Arg Lys Asp Ser Arg Gly
290 295
<210> 543
<211> 310
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 20 - бета нативный
<400> 543
Met Leu Leu Leu Leu Leu Leu Leu Gly Pro Gly Ser Gly Leu Gly Ala
1 5 10 15
Val Val Ser Gln His Pro Ser Arg Val Ile Cys Lys Ser Gly Thr Ser
20 25 30
Val Lys Ile Glu Cys Arg Ser Leu Asp Phe Gln Ala Thr Thr Met Phe
35 40 45
Trp Tyr Arg Gln Phe Pro Lys Gln Ser Leu Met Leu Met Ala Thr Ser
50 55 60
Asn Glu Gly Ser Lys Ala Thr Tyr Glu Gln Gly Val Glu Lys Asp Lys
65 70 75 80
Phe Leu Ile Asn His Ala Ser Leu Thr Leu Ser Thr Leu Thr Val Thr
85 90 95
Ser Ala His Pro Glu Asp Ser Ser Phe Tyr Ile Cys Ser Ala Ser Ser
100 105 110
Leu Ala Arg Arg Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu
115 120 125
Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val
130 135 140
Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu
145 150 155 160
Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp
165 170 175
Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln
180 185 190
Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser
195 200 205
Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His
210 215 220
Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp
225 230 235 240
Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala
245 250 255
Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly
260 265 270
Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr
275 280 285
Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys
290 295 300
Arg Lys Asp Ser Arg Gly
305 310
<210> 544
<211> 310
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 20 - бета, цистеин-модифицированный
<400> 544
Met Leu Leu Leu Leu Leu Leu Leu Gly Pro Gly Ser Gly Leu Gly Ala
1 5 10 15
Val Val Ser Gln His Pro Ser Arg Val Ile Cys Lys Ser Gly Thr Ser
20 25 30
Val Lys Ile Glu Cys Arg Ser Leu Asp Phe Gln Ala Thr Thr Met Phe
35 40 45
Trp Tyr Arg Gln Phe Pro Lys Gln Ser Leu Met Leu Met Ala Thr Ser
50 55 60
Asn Glu Gly Ser Lys Ala Thr Tyr Glu Gln Gly Val Glu Lys Asp Lys
65 70 75 80
Phe Leu Ile Asn His Ala Ser Leu Thr Leu Ser Thr Leu Thr Val Thr
85 90 95
Ser Ala His Pro Glu Asp Ser Ser Phe Tyr Ile Cys Ser Ala Ser Ser
100 105 110
Leu Ala Arg Arg Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu
115 120 125
Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val
130 135 140
Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu
145 150 155 160
Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp
165 170 175
Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln
180 185 190
Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser
195 200 205
Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His
210 215 220
Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp
225 230 235 240
Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala
245 250 255
Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly
260 265 270
Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr
275 280 285
Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys
290 295 300
Arg Lys Asp Ser Arg Gly
305 310
<210> 545
<211> 117
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 20 бета, вариабельная область
<400> 545
Gly Ala Val Val Ser Gln His Pro Ser Arg Val Ile Cys Lys Ser Gly
1 5 10 15
Thr Ser Val Lys Ile Glu Cys Arg Ser Leu Asp Phe Gln Ala Thr Thr
20 25 30
Met Phe Trp Tyr Arg Gln Phe Pro Lys Gln Ser Leu Met Leu Met Ala
35 40 45
Thr Ser Asn Glu Gly Ser Lys Ala Thr Tyr Glu Gln Gly Val Glu Lys
50 55 60
Asp Lys Phe Leu Ile Asn His Ala Ser Leu Thr Leu Ser Thr Leu Thr
65 70 75 80
Val Thr Ser Ala His Pro Glu Asp Ser Ser Phe Tyr Ile Cys Ser Ala
85 90 95
Ser Ser Leu Ala Arg Arg Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr
100 105 110
Arg Leu Thr Val Thr
115
<210> 546
<211> 6
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 20 бета CDR1
<400> 546
Asp Phe Gln Ala Thr Thr
1 5
<210> 547
<211> 7
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 20 бета CDR2
<400> 547
Ser Asn Glu Gly Ser Lys Ala
1 5
<210> 548
<211> 13
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 20 бета CDR3
<400> 548
Ser Ala Ser Ser Leu Ala Arg Arg Ser Tyr Glu Gln Tyr
1 5 10
<210> 549
<211> 14
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 20 бета, Сигнальный пептид
<400> 549
Met Leu Leu Leu Leu Leu Leu Leu Gly Pro Gly Ser Gly Leu
1 5 10
<210> 550
<211> 259
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 21 - альфа нативный
<400> 550
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
1 5 10 15
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln Ser Tyr
20 25 30
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
35 40 45
Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly Arg Tyr
50 55 60
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
65 70 75 80
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Arg Glu
85 90 95
Gly Arg Val Thr Gly Gly Gly Asn Lys Leu Thr Phe Gly Thr Gly Thr
100 105 110
Gln Leu Lys Val Glu Leu Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr
115 120 125
Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr
130 135 140
Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val
145 150 155 160
Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys
165 170 175
Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala
180 185 190
Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser
195 200 205
Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr
210 215 220
Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile
225 230 235 240
Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu
245 250 255
Trp Ser Ser
<210> 551
<211> 259
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 21 - альфа, цистеин-модифицированный
<400> 551
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
1 5 10 15
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln Ser Tyr
20 25 30
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
35 40 45
Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly Arg Tyr
50 55 60
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
65 70 75 80
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Arg Glu
85 90 95
Gly Arg Val Thr Gly Gly Gly Asn Lys Leu Thr Phe Gly Thr Gly Thr
100 105 110
Gln Leu Lys Val Glu Leu Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr
115 120 125
Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr
130 135 140
Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val
145 150 155 160
Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys
165 170 175
Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala
180 185 190
Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser
195 200 205
Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr
210 215 220
Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile
225 230 235 240
Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu
245 250 255
Trp Ser Ser
<210> 552
<211> 279
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 21 - альфа нативный
<400> 552
Met Ser Leu Ser Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu
1 5 10 15
Gly Pro Gly Ile Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe
20 25 30
Val Gln Glu Lys Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser
35 40 45
Asp Gln Ser Tyr Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu
50 55 60
Met Ile Phe Leu Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr
65 70 75 80
Glu Gly Arg Tyr Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn
85 90 95
Leu Val Ile Ser Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys
100 105 110
Ala Met Arg Glu Gly Arg Val Thr Gly Gly Gly Asn Lys Leu Thr Phe
115 120 125
Gly Thr Gly Thr Gln Leu Lys Val Glu Leu Asn Ile Gln Asn Pro Asp
130 135 140
Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val
145 150 155 160
Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys
165 170 175
Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser
180 185 190
Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp
195 200 205
Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr
210 215 220
Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys
225 230 235 240
Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile
245 250 255
Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met
260 265 270
Thr Leu Arg Leu Trp Ser Ser
275
<210> 553
<211> 279
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 21 - альфа, цистеин-модифицированный
<400> 553
Met Ser Leu Ser Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu
1 5 10 15
Gly Pro Gly Ile Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe
20 25 30
Val Gln Glu Lys Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser
35 40 45
Asp Gln Ser Tyr Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu
50 55 60
Met Ile Phe Leu Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr
65 70 75 80
Glu Gly Arg Tyr Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn
85 90 95
Leu Val Ile Ser Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys
100 105 110
Ala Met Arg Glu Gly Arg Val Thr Gly Gly Gly Asn Lys Leu Thr Phe
115 120 125
Gly Thr Gly Thr Gln Leu Lys Val Glu Leu Asn Ile Gln Asn Pro Asp
130 135 140
Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val
145 150 155 160
Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys
165 170 175
Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser
180 185 190
Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp
195 200 205
Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr
210 215 220
Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys
225 230 235 240
Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile
245 250 255
Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met
260 265 270
Thr Leu Arg Leu Trp Ser Ser
275
<210> 554
<211> 118
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 21 альфа, вариабельная область
<400> 554
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
1 5 10 15
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln Ser Tyr
20 25 30
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
35 40 45
Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly Arg Tyr
50 55 60
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
65 70 75 80
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Arg Glu
85 90 95
Gly Arg Val Thr Gly Gly Gly Asn Lys Leu Thr Phe Gly Thr Gly Thr
100 105 110
Gln Leu Lys Val Glu Leu
115
<210> 555
<211> 15
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 21 альфа CDR3
<400> 555
Ala Met Arg Glu Gly Arg Val Thr Gly Gly Gly Asn Lys Leu Thr
1 5 10 15
<210> 556
<211> 291
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 21 - бета нативный
<400> 556
Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Gln Val Leu Lys Thr Gly
1 5 10 15
Gln Ser Met Thr Leu Gln Cys Ala Gln Asp Met Asn His Asn Ser Met
20 25 30
Tyr Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu Ile Tyr Tyr
35 40 45
Ser Ala Ser Glu Gly Thr Thr Asp Lys Gly Glu Val Pro Asn Gly Tyr
50 55 60
Asn Val Ser Arg Leu Asn Lys Arg Glu Phe Ser Leu Arg Leu Glu Ser
65 70 75 80
Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser Arg Pro Trp
85 90 95
Gly Asn Gln Asn Thr Glu Ala Phe Phe Gly Gln Gly Thr Arg Leu Thr
100 105 110
Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val Phe
115 120 125
Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val
130 135 140
Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp Trp
145 150 155 160
Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro
165 170 175
Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser
180 185 190
Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe
195 200 205
Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr
210 215 220
Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp
225 230 235 240
Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly Val
245 250 255
Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu
260 265 270
Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg
275 280 285
Lys Asp Phe
290
<210> 557
<211> 291
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 21 - бета, цистеин-модифицированный
<400> 557
Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Gln Val Leu Lys Thr Gly
1 5 10 15
Gln Ser Met Thr Leu Gln Cys Ala Gln Asp Met Asn His Asn Ser Met
20 25 30
Tyr Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu Ile Tyr Tyr
35 40 45
Ser Ala Ser Glu Gly Thr Thr Asp Lys Gly Glu Val Pro Asn Gly Tyr
50 55 60
Asn Val Ser Arg Leu Asn Lys Arg Glu Phe Ser Leu Arg Leu Glu Ser
65 70 75 80
Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser Arg Pro Trp
85 90 95
Gly Asn Gln Asn Thr Glu Ala Phe Phe Gly Gln Gly Thr Arg Leu Thr
100 105 110
Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val Phe
115 120 125
Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val
130 135 140
Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp Trp
145 150 155 160
Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro
165 170 175
Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser
180 185 190
Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe
195 200 205
Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr
210 215 220
Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp
225 230 235 240
Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly Val
245 250 255
Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu
260 265 270
Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg
275 280 285
Lys Asp Phe
290
<210> 558
<211> 324
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 21 - бета нативный
<400> 558
Met His Arg Pro Arg Arg Pro Leu His Pro Val Ala Pro Ala Met Ser
1 5 10 15
Ile Gly Leu Leu Cys Cys Val Ala Phe Ser Leu Leu Trp Ala Ser Pro
20 25 30
Val Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Gln Val Leu Lys Thr
35 40 45
Gly Gln Ser Met Thr Leu Gln Cys Ala Gln Asp Met Asn His Asn Ser
50 55 60
Met Tyr Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu Ile Tyr
65 70 75 80
Tyr Ser Ala Ser Glu Gly Thr Thr Asp Lys Gly Glu Val Pro Asn Gly
85 90 95
Tyr Asn Val Ser Arg Leu Asn Lys Arg Glu Phe Ser Leu Arg Leu Glu
100 105 110
Ser Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser Arg Pro
115 120 125
Trp Gly Asn Gln Asn Thr Glu Ala Phe Phe Gly Gln Gly Thr Arg Leu
130 135 140
Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val
145 150 155 160
Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu
165 170 175
Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp
180 185 190
Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln
195 200 205
Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser
210 215 220
Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His
225 230 235 240
Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp
245 250 255
Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala
260 265 270
Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly
275 280 285
Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr
290 295 300
Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys
305 310 315 320
Arg Lys Asp Phe
<210> 559
<211> 324
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 21 - бета, цистеин-модифицированный
<400> 559
Met His Arg Pro Arg Arg Pro Leu His Pro Val Ala Pro Ala Met Ser
1 5 10 15
Ile Gly Leu Leu Cys Cys Val Ala Phe Ser Leu Leu Trp Ala Ser Pro
20 25 30
Val Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Gln Val Leu Lys Thr
35 40 45
Gly Gln Ser Met Thr Leu Gln Cys Ala Gln Asp Met Asn His Asn Ser
50 55 60
Met Tyr Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu Ile Tyr
65 70 75 80
Tyr Ser Ala Ser Glu Gly Thr Thr Asp Lys Gly Glu Val Pro Asn Gly
85 90 95
Tyr Asn Val Ser Arg Leu Asn Lys Arg Glu Phe Ser Leu Arg Leu Glu
100 105 110
Ser Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser Arg Pro
115 120 125
Trp Gly Asn Gln Asn Thr Glu Ala Phe Phe Gly Gln Gly Thr Arg Leu
130 135 140
Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val
145 150 155 160
Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu
165 170 175
Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp
180 185 190
Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln
195 200 205
Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser
210 215 220
Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His
225 230 235 240
Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp
245 250 255
Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala
260 265 270
Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly
275 280 285
Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr
290 295 300
Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys
305 310 315 320
Arg Lys Asp Phe
<210> 560
<211> 114
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 21 бета, вариабельная область
<400> 560
Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Gln Val Leu Lys Thr Gly
1 5 10 15
Gln Ser Met Thr Leu Gln Cys Ala Gln Asp Met Asn His Asn Ser Met
20 25 30
Tyr Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu Ile Tyr Tyr
35 40 45
Ser Ala Ser Glu Gly Thr Thr Asp Lys Gly Glu Val Pro Asn Gly Tyr
50 55 60
Asn Val Ser Arg Leu Asn Lys Arg Glu Phe Ser Leu Arg Leu Glu Ser
65 70 75 80
Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser Arg Pro Trp
85 90 95
Gly Asn Gln Asn Thr Glu Ala Phe Phe Gly Gln Gly Thr Arg Leu Thr
100 105 110
Val Val
<210> 561
<211> 5
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 21/ TCR 27 бета CDR1
<400> 561
Met Asn His Asn Ser
1 5
<210> 562
<211> 6
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 21/ TCR 27 бета CDR2
<400> 562
Ser Ala Ser Glu Gly Thr
1 5
<210> 563
<211> 13
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 21 бета CDR3
<400> 563
Ala Ser Arg Pro Trp Gly Asn Gln Asn Thr Glu Ala Phe
1 5 10
<210> 564
<211> 33
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 21/ TCR 27 бета, Сигнальный пептид
<400> 564
Met His Arg Pro Arg Arg Pro Leu His Pro Val Ala Pro Ala Met Ser
1 5 10 15
Ile Gly Leu Leu Cys Cys Val Ala Phe Ser Leu Leu Trp Ala Ser Pro
20 25 30
Val
<210> 565
<211> 253
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 22 - альфа нативный
<400> 565
Gly Gln Asn Ile Asp Gln Pro Thr Glu Met Thr Ala Thr Glu Gly Ala
1 5 10 15
Ile Val Gln Ile Asn Cys Thr Tyr Gln Thr Ser Gly Phe Asn Gly Leu
20 25 30
Phe Trp Tyr Gln Gln His Ala Gly Glu Ala Pro Thr Phe Leu Ser Tyr
35 40 45
Asn Val Leu Asp Gly Leu Glu Glu Lys Gly Arg Phe Ser Ser Phe Leu
50 55 60
Ser Arg Ser Lys Gly Tyr Ser Tyr Leu Leu Leu Lys Glu Leu Gln Met
65 70 75 80
Lys Asp Ser Ala Ser Tyr Leu Cys Ala Val Arg Ala Arg Met Asp Ser
85 90 95
Asn Tyr Gln Leu Ile Trp Gly Ala Gly Thr Lys Leu Ile Ile Lys Pro
100 105 110
Asp Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys
115 120 125
Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr
130 135 140
Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr
145 150 155 160
Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala
165 170 175
Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser
180 185 190
Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp
195 200 205
Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe
210 215 220
Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala
225 230 235 240
Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 566
<211> 253
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 22 - альфа, цистеин-модифицированный
<400> 566
Gly Gln Asn Ile Asp Gln Pro Thr Glu Met Thr Ala Thr Glu Gly Ala
1 5 10 15
Ile Val Gln Ile Asn Cys Thr Tyr Gln Thr Ser Gly Phe Asn Gly Leu
20 25 30
Phe Trp Tyr Gln Gln His Ala Gly Glu Ala Pro Thr Phe Leu Ser Tyr
35 40 45
Asn Val Leu Asp Gly Leu Glu Glu Lys Gly Arg Phe Ser Ser Phe Leu
50 55 60
Ser Arg Ser Lys Gly Tyr Ser Tyr Leu Leu Leu Lys Glu Leu Gln Met
65 70 75 80
Lys Asp Ser Ala Ser Tyr Leu Cys Ala Val Arg Ala Arg Met Asp Ser
85 90 95
Asn Tyr Gln Leu Ile Trp Gly Ala Gly Thr Lys Leu Ile Ile Lys Pro
100 105 110
Asp Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys
115 120 125
Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr
130 135 140
Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys
145 150 155 160
Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala
165 170 175
Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser
180 185 190
Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp
195 200 205
Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe
210 215 220
Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala
225 230 235 240
Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 567
<211> 287
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 22 - альфа нативный
<400> 567
Met Ala Gln Glu Leu Gly Met Gln Cys Gln Ala Arg Gly Ile Leu Gln
1 5 10 15
Gln Met Trp Gly Val Phe Leu Leu Tyr Val Ser Met Lys Met Gly Gly
20 25 30
Thr Thr Gly Gln Asn Ile Asp Gln Pro Thr Glu Met Thr Ala Thr Glu
35 40 45
Gly Ala Ile Val Gln Ile Asn Cys Thr Tyr Gln Thr Ser Gly Phe Asn
50 55 60
Gly Leu Phe Trp Tyr Gln Gln His Ala Gly Glu Ala Pro Thr Phe Leu
65 70 75 80
Ser Tyr Asn Val Leu Asp Gly Leu Glu Glu Lys Gly Arg Phe Ser Ser
85 90 95
Phe Leu Ser Arg Ser Lys Gly Tyr Ser Tyr Leu Leu Leu Lys Glu Leu
100 105 110
Gln Met Lys Asp Ser Ala Ser Tyr Leu Cys Ala Val Arg Ala Arg Met
115 120 125
Asp Ser Asn Tyr Gln Leu Ile Trp Gly Ala Gly Thr Lys Leu Ile Ile
130 135 140
Lys Pro Asp Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp
145 150 155 160
Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser
165 170 175
Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp
180 185 190
Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala
195 200 205
Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn
210 215 220
Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser
225 230 235 240
Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu
245 250 255
Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys
260 265 270
Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
275 280 285
<210> 568
<211> 287
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 22 - альфа, цистеин-модифицированный
<400> 568
Met Ala Gln Glu Leu Gly Met Gln Cys Gln Ala Arg Gly Ile Leu Gln
1 5 10 15
Gln Met Trp Gly Val Phe Leu Leu Tyr Val Ser Met Lys Met Gly Gly
20 25 30
Thr Thr Gly Gln Asn Ile Asp Gln Pro Thr Glu Met Thr Ala Thr Glu
35 40 45
Gly Ala Ile Val Gln Ile Asn Cys Thr Tyr Gln Thr Ser Gly Phe Asn
50 55 60
Gly Leu Phe Trp Tyr Gln Gln His Ala Gly Glu Ala Pro Thr Phe Leu
65 70 75 80
Ser Tyr Asn Val Leu Asp Gly Leu Glu Glu Lys Gly Arg Phe Ser Ser
85 90 95
Phe Leu Ser Arg Ser Lys Gly Tyr Ser Tyr Leu Leu Leu Lys Glu Leu
100 105 110
Gln Met Lys Asp Ser Ala Ser Tyr Leu Cys Ala Val Arg Ala Arg Met
115 120 125
Asp Ser Asn Tyr Gln Leu Ile Trp Gly Ala Gly Thr Lys Leu Ile Ile
130 135 140
Lys Pro Asp Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp
145 150 155 160
Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser
165 170 175
Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp
180 185 190
Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala
195 200 205
Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn
210 215 220
Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser
225 230 235 240
Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu
245 250 255
Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys
260 265 270
Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
275 280 285
<210> 569
<211> 112
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 22 альфа, вариабельная область
<400> 569
Gly Gln Asn Ile Asp Gln Pro Thr Glu Met Thr Ala Thr Glu Gly Ala
1 5 10 15
Ile Val Gln Ile Asn Cys Thr Tyr Gln Thr Ser Gly Phe Asn Gly Leu
20 25 30
Phe Trp Tyr Gln Gln His Ala Gly Glu Ala Pro Thr Phe Leu Ser Tyr
35 40 45
Asn Val Leu Asp Gly Leu Glu Glu Lys Gly Arg Phe Ser Ser Phe Leu
50 55 60
Ser Arg Ser Lys Gly Tyr Ser Tyr Leu Leu Leu Lys Glu Leu Gln Met
65 70 75 80
Lys Asp Ser Ala Ser Tyr Leu Cys Ala Val Arg Ala Arg Met Asp Ser
85 90 95
Asn Tyr Gln Leu Ile Trp Gly Ala Gly Thr Lys Leu Ile Ile Lys Pro
100 105 110
<210> 570
<211> 6
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 22/ TCR 44 альфа CDR1
<400> 570
Thr Ser Gly Phe Asn Gly
1 5
<210> 571
<211> 6
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 22/ TCR 44 альфа CDR2
<400> 571
Asn Val Leu Asp Gly Leu
1 5
<210> 572
<211> 13
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 22 альфа CDR3
<400> 572
Ala Val Arg Ala Arg Met Asp Ser Asn Tyr Gln Leu Ile
1 5 10
<210> 573
<211> 34
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 22/ TCR 44альфа, Сигнальный пептид
<400> 573
Met Ala Gln Glu Leu Gly Met Gln Cys Gln Ala Arg Gly Ile Leu Gln
1 5 10 15
Gln Met Trp Gly Val Phe Leu Leu Tyr Val Ser Met Lys Met Gly Gly
20 25 30
Thr Thr
<210> 574
<211> 289
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 22 - бета нативный
<400> 574
Asp Ala Gly Val Thr Gln Ser Pro Thr His Leu Ile Lys Thr Arg Gly
1 5 10 15
Gln His Val Thr Leu Arg Cys Ser Pro Ile Ser Gly His Lys Ser Val
20 25 30
Ser Trp Tyr Gln Gln Val Leu Gly Gln Gly Pro Gln Phe Ile Phe Gln
35 40 45
Tyr Tyr Glu Lys Glu Glu Arg Gly Arg Gly Asn Phe Pro Asp Arg Phe
50 55 60
Ser Ala Arg Gln Phe Pro Asn Tyr Ser Ser Glu Leu Asn Val Asn Ala
65 70 75 80
Leu Leu Leu Gly Asp Ser Ala Leu Tyr Leu Cys Ala Ser Ser Arg Thr
85 90 95
Glu Asn Tyr Gly Tyr Thr Phe Gly Ser Gly Thr Arg Leu Thr Val Val
100 105 110
Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val Phe Glu Pro
115 120 125
Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys Leu
130 135 140
Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp Trp Val Asn
145 150 155 160
Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro Leu Lys
165 170 175
Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg Leu
180 185 190
Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg Cys
195 200 205
Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln Asp
210 215 220
Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly Arg
225 230 235 240
Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly Val Leu Ser
245 250 255
Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr Ala
260 265 270
Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys Asp
275 280 285
Phe
<210> 575
<211> 289
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 22 - бета, цистеин-модифицированный
<400> 575
Asp Ala Gly Val Thr Gln Ser Pro Thr His Leu Ile Lys Thr Arg Gly
1 5 10 15
Gln His Val Thr Leu Arg Cys Ser Pro Ile Ser Gly His Lys Ser Val
20 25 30
Ser Trp Tyr Gln Gln Val Leu Gly Gln Gly Pro Gln Phe Ile Phe Gln
35 40 45
Tyr Tyr Glu Lys Glu Glu Arg Gly Arg Gly Asn Phe Pro Asp Arg Phe
50 55 60
Ser Ala Arg Gln Phe Pro Asn Tyr Ser Ser Glu Leu Asn Val Asn Ala
65 70 75 80
Leu Leu Leu Gly Asp Ser Ala Leu Tyr Leu Cys Ala Ser Ser Arg Thr
85 90 95
Glu Asn Tyr Gly Tyr Thr Phe Gly Ser Gly Thr Arg Leu Thr Val Val
100 105 110
Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val Phe Glu Pro
115 120 125
Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys Leu
130 135 140
Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp Trp Val Asn
145 150 155 160
Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro Leu Lys
165 170 175
Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg Leu
180 185 190
Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg Cys
195 200 205
Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln Asp
210 215 220
Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly Arg
225 230 235 240
Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly Val Leu Ser
245 250 255
Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr Ala
260 265 270
Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys Asp
275 280 285
Phe
<210> 576
<211> 308
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 22 - бета нативный
<400> 576
Met Gly Pro Gly Leu Leu Cys Trp Val Leu Leu Cys Leu Leu Gly Ala
1 5 10 15
Gly Pro Val Asp Ala Gly Val Thr Gln Ser Pro Thr His Leu Ile Lys
20 25 30
Thr Arg Gly Gln His Val Thr Leu Arg Cys Ser Pro Ile Ser Gly His
35 40 45
Lys Ser Val Ser Trp Tyr Gln Gln Val Leu Gly Gln Gly Pro Gln Phe
50 55 60
Ile Phe Gln Tyr Tyr Glu Lys Glu Glu Arg Gly Arg Gly Asn Phe Pro
65 70 75 80
Asp Arg Phe Ser Ala Arg Gln Phe Pro Asn Tyr Ser Ser Glu Leu Asn
85 90 95
Val Asn Ala Leu Leu Leu Gly Asp Ser Ala Leu Tyr Leu Cys Ala Ser
100 105 110
Ser Arg Thr Glu Asn Tyr Gly Tyr Thr Phe Gly Ser Gly Thr Arg Leu
115 120 125
Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val
130 135 140
Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu
145 150 155 160
Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp
165 170 175
Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln
180 185 190
Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser
195 200 205
Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His
210 215 220
Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp
225 230 235 240
Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala
245 250 255
Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly
260 265 270
Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr
275 280 285
Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys
290 295 300
Arg Lys Asp Phe
305
<210> 577
<211> 308
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 22 - бета, цистеин-модифицированный
<400> 577
Met Gly Pro Gly Leu Leu Cys Trp Val Leu Leu Cys Leu Leu Gly Ala
1 5 10 15
Gly Pro Val Asp Ala Gly Val Thr Gln Ser Pro Thr His Leu Ile Lys
20 25 30
Thr Arg Gly Gln His Val Thr Leu Arg Cys Ser Pro Ile Ser Gly His
35 40 45
Lys Ser Val Ser Trp Tyr Gln Gln Val Leu Gly Gln Gly Pro Gln Phe
50 55 60
Ile Phe Gln Tyr Tyr Glu Lys Glu Glu Arg Gly Arg Gly Asn Phe Pro
65 70 75 80
Asp Arg Phe Ser Ala Arg Gln Phe Pro Asn Tyr Ser Ser Glu Leu Asn
85 90 95
Val Asn Ala Leu Leu Leu Gly Asp Ser Ala Leu Tyr Leu Cys Ala Ser
100 105 110
Ser Arg Thr Glu Asn Tyr Gly Tyr Thr Phe Gly Ser Gly Thr Arg Leu
115 120 125
Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val
130 135 140
Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu
145 150 155 160
Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp
165 170 175
Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln
180 185 190
Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser
195 200 205
Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His
210 215 220
Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp
225 230 235 240
Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala
245 250 255
Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly
260 265 270
Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr
275 280 285
Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys
290 295 300
Arg Lys Asp Phe
305
<210> 578
<211> 112
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 22 бета, вариабельная область
<400> 578
Asp Ala Gly Val Thr Gln Ser Pro Thr His Leu Ile Lys Thr Arg Gly
1 5 10 15
Gln His Val Thr Leu Arg Cys Ser Pro Ile Ser Gly His Lys Ser Val
20 25 30
Ser Trp Tyr Gln Gln Val Leu Gly Gln Gly Pro Gln Phe Ile Phe Gln
35 40 45
Tyr Tyr Glu Lys Glu Glu Arg Gly Arg Gly Asn Phe Pro Asp Arg Phe
50 55 60
Ser Ala Arg Gln Phe Pro Asn Tyr Ser Ser Glu Leu Asn Val Asn Ala
65 70 75 80
Leu Leu Leu Gly Asp Ser Ala Leu Tyr Leu Cys Ala Ser Ser Arg Thr
85 90 95
Glu Asn Tyr Gly Tyr Thr Phe Gly Ser Gly Thr Arg Leu Thr Val Val
100 105 110
<210> 579
<211> 5
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 22 бета CDR1
<400> 579
Ser Gly His Lys Ser
1 5
<210> 580
<211> 6
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 22 бета CDR2
<400> 580
Tyr Tyr Glu Lys Glu Glu
1 5
<210> 581
<211> 11
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 22 бета CDR3
<400> 581
Ala Ser Ser Arg Thr Glu Asn Tyr Gly Tyr Thr
1 5 10
<210> 582
<211> 19
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 22 бета, Сигнальный пептид
<400> 582
Met Gly Pro Gly Leu Leu Cys Trp Val Leu Leu Cys Leu Leu Gly Ala
1 5 10 15
Gly Pro Val
<210> 583
<211> 257
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 23 - альфа нативный
<400> 583
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
1 5 10 15
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln Ser Tyr
20 25 30
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
35 40 45
Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly Arg Tyr
50 55 60
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
65 70 75 80
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Ser Pro
85 90 95
Pro Gly Gly Ser Ala Arg Gln Leu Thr Phe Gly Ser Gly Thr Gln Leu
100 105 110
Thr Val Leu Pro Asp Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu
115 120 125
Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe
130 135 140
Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile
145 150 155 160
Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn
165 170 175
Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala
180 185 190
Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu
195 200 205
Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr
210 215 220
Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu
225 230 235 240
Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser
245 250 255
Ser
<210> 584
<211> 257
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 23 - альфа, цистеин-модифицированный
<400> 584
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
1 5 10 15
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln Ser Tyr
20 25 30
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
35 40 45
Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly Arg Tyr
50 55 60
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
65 70 75 80
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Ser Pro
85 90 95
Pro Gly Gly Ser Ala Arg Gln Leu Thr Phe Gly Ser Gly Thr Gln Leu
100 105 110
Thr Val Leu Pro Asp Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu
115 120 125
Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe
130 135 140
Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile
145 150 155 160
Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn
165 170 175
Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala
180 185 190
Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu
195 200 205
Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr
210 215 220
Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu
225 230 235 240
Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser
245 250 255
Ser
<210> 585
<211> 277
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 23 - альфа нативный
<400> 585
Met Ser Leu Ser Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu
1 5 10 15
Gly Pro Gly Ile Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe
20 25 30
Val Gln Glu Lys Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser
35 40 45
Asp Gln Ser Tyr Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu
50 55 60
Met Ile Phe Leu Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr
65 70 75 80
Glu Gly Arg Tyr Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn
85 90 95
Leu Val Ile Ser Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys
100 105 110
Ala Met Ser Pro Pro Gly Gly Ser Ala Arg Gln Leu Thr Phe Gly Ser
115 120 125
Gly Thr Gln Leu Thr Val Leu Pro Asp Ile Gln Asn Pro Asp Pro Ala
130 135 140
Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu
145 150 155 160
Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser
165 170 175
Asp Val Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp
180 185 190
Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala
195 200 205
Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe
210 215 220
Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe
225 230 235 240
Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe
245 250 255
Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu
260 265 270
Arg Leu Trp Ser Ser
275
<210> 586
<211> 277
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 23 - альфа, цистеин-модифицированный
<400> 586
Met Ser Leu Ser Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu
1 5 10 15
Gly Pro Gly Ile Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe
20 25 30
Val Gln Glu Lys Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser
35 40 45
Asp Gln Ser Tyr Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu
50 55 60
Met Ile Phe Leu Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr
65 70 75 80
Glu Gly Arg Tyr Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn
85 90 95
Leu Val Ile Ser Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys
100 105 110
Ala Met Ser Pro Pro Gly Gly Ser Ala Arg Gln Leu Thr Phe Gly Ser
115 120 125
Gly Thr Gln Leu Thr Val Leu Pro Asp Ile Gln Asn Pro Asp Pro Ala
130 135 140
Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu
145 150 155 160
Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser
165 170 175
Asp Val Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp
180 185 190
Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala
195 200 205
Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe
210 215 220
Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe
225 230 235 240
Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe
245 250 255
Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu
260 265 270
Arg Leu Trp Ser Ser
275
<210> 587
<211> 116
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 23 альфа, вариабельная область
<400> 587
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
1 5 10 15
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln Ser Tyr
20 25 30
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
35 40 45
Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly Arg Tyr
50 55 60
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
65 70 75 80
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Ser Pro
85 90 95
Pro Gly Gly Ser Ala Arg Gln Leu Thr Phe Gly Ser Gly Thr Gln Leu
100 105 110
Thr Val Leu Pro
115
<210> 588
<211> 13
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 23 альфа CDR3
<400> 588
Ala Met Ser Pro Pro Gly Gly Ser Ala Arg Gln Leu Thr
1 5 10
<210> 589
<211> 291
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 23 - бета нативный
<400> 589
Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys Arg Thr Gly
1 5 10 15
Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His Glu Asn Met
20 25 30
Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu Ile Tyr Phe
35 40 45
Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro Glu Gly Tyr
50 55 60
Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile Leu Glu Ser
65 70 75 80
Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser Ser Pro Trp
85 90 95
Gly Gln Ser Asn Gln Pro Gln His Phe Gly Asp Gly Thr Arg Leu Ser
100 105 110
Ile Leu Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val Phe
115 120 125
Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val
130 135 140
Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp Trp
145 150 155 160
Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro
165 170 175
Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser
180 185 190
Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe
195 200 205
Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr
210 215 220
Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp
225 230 235 240
Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly Val
245 250 255
Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu
260 265 270
Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg
275 280 285
Lys Asp Phe
290
<210> 590
<211> 291
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 23 - бета, цистеин-модифицированный
<400> 590
Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys Arg Thr Gly
1 5 10 15
Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His Glu Asn Met
20 25 30
Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu Ile Tyr Phe
35 40 45
Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro Glu Gly Tyr
50 55 60
Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile Leu Glu Ser
65 70 75 80
Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser Ser Pro Trp
85 90 95
Gly Gln Ser Asn Gln Pro Gln His Phe Gly Asp Gly Thr Arg Leu Ser
100 105 110
Ile Leu Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val Phe
115 120 125
Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val
130 135 140
Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp Trp
145 150 155 160
Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro
165 170 175
Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser
180 185 190
Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe
195 200 205
Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr
210 215 220
Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp
225 230 235 240
Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly Val
245 250 255
Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu
260 265 270
Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg
275 280 285
Lys Asp Phe
290
<210> 591
<211> 310
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 23 - бета нативный
<400> 591
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Ser Pro Trp Gly Gln Ser Asn Gln Pro Gln His Phe Gly Asp Gly Thr
115 120 125
Arg Leu Ser Ile Leu Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe
305 310
<210> 592
<211> 310
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 23 - бета, цистеин-модифицированный
<400> 592
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Ser Pro Trp Gly Gln Ser Asn Gln Pro Gln His Phe Gly Asp Gly Thr
115 120 125
Arg Leu Ser Ile Leu Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe
305 310
<210> 593
<211> 114
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 23 бета, вариабельная область
<400> 593
Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys Arg Thr Gly
1 5 10 15
Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His Glu Asn Met
20 25 30
Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu Ile Tyr Phe
35 40 45
Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro Glu Gly Tyr
50 55 60
Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile Leu Glu Ser
65 70 75 80
Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser Ser Pro Trp
85 90 95
Gly Gln Ser Asn Gln Pro Gln His Phe Gly Asp Gly Thr Arg Leu Ser
100 105 110
Ile Leu
<210> 594
<211> 13
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 23 бета CDR3
<400> 594
Ala Ser Ser Pro Trp Gly Gln Ser Asn Gln Pro Gln His
1 5 10
<210> 595
<211> 258
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 24 - альфа нативный
<400> 595
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
1 5 10 15
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln Ser Tyr
20 25 30
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
35 40 45
Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly Arg Tyr
50 55 60
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
65 70 75 80
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Arg Glu
85 90 95
Gly Arg Gly Asp Ser Trp Gly Lys Leu Gln Phe Gly Ala Gly Thr Gln
100 105 110
Val Val Val Thr Pro Asp Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln
115 120 125
Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp
130 135 140
Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr
145 150 155 160
Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser
165 170 175
Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn
180 185 190
Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro
195 200 205
Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp
210 215 220
Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu
225 230 235 240
Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp
245 250 255
Ser Ser
<210> 596
<211> 258
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 24 - альфа, цистеин-модифицированный
<400> 596
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
1 5 10 15
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln Ser Tyr
20 25 30
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
35 40 45
Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly Arg Tyr
50 55 60
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
65 70 75 80
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Arg Glu
85 90 95
Gly Arg Gly Asp Ser Trp Gly Lys Leu Gln Phe Gly Ala Gly Thr Gln
100 105 110
Val Val Val Thr Pro Asp Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln
115 120 125
Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp
130 135 140
Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr
145 150 155 160
Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser
165 170 175
Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn
180 185 190
Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro
195 200 205
Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp
210 215 220
Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu
225 230 235 240
Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp
245 250 255
Ser Ser
<210> 597
<211> 278
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 24 - альфа нативный
<400> 597
Met Ser Leu Ser Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu
1 5 10 15
Gly Pro Gly Ile Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe
20 25 30
Val Gln Glu Lys Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser
35 40 45
Asp Gln Ser Tyr Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu
50 55 60
Met Ile Phe Leu Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr
65 70 75 80
Glu Gly Arg Tyr Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn
85 90 95
Leu Val Ile Ser Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys
100 105 110
Ala Met Arg Glu Gly Arg Gly Asp Ser Trp Gly Lys Leu Gln Phe Gly
115 120 125
Ala Gly Thr Gln Val Val Val Thr Pro Asp Ile Gln Asn Pro Asp Pro
130 135 140
Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys
145 150 155 160
Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp
165 170 175
Ser Asp Val Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met
180 185 190
Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe
195 200 205
Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe
210 215 220
Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser
225 230 235 240
Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly
245 250 255
Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr
260 265 270
Leu Arg Leu Trp Ser Ser
275
<210> 598
<211> 278
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 24 - альфа, цистеин-модифицированный
<400> 598
Met Ser Leu Ser Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu
1 5 10 15
Gly Pro Gly Ile Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe
20 25 30
Val Gln Glu Lys Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser
35 40 45
Asp Gln Ser Tyr Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu
50 55 60
Met Ile Phe Leu Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr
65 70 75 80
Glu Gly Arg Tyr Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn
85 90 95
Leu Val Ile Ser Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys
100 105 110
Ala Met Arg Glu Gly Arg Gly Asp Ser Trp Gly Lys Leu Gln Phe Gly
115 120 125
Ala Gly Thr Gln Val Val Val Thr Pro Asp Ile Gln Asn Pro Asp Pro
130 135 140
Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys
145 150 155 160
Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp
165 170 175
Ser Asp Val Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met
180 185 190
Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe
195 200 205
Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe
210 215 220
Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser
225 230 235 240
Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly
245 250 255
Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr
260 265 270
Leu Arg Leu Trp Ser Ser
275
<210> 599
<211> 117
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 24 альфа, вариабельная область
<400> 599
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
1 5 10 15
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln Ser Tyr
20 25 30
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
35 40 45
Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly Arg Tyr
50 55 60
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
65 70 75 80
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Arg Glu
85 90 95
Gly Arg Gly Asp Ser Trp Gly Lys Leu Gln Phe Gly Ala Gly Thr Gln
100 105 110
Val Val Val Thr Pro
115
<210> 600
<211> 14
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 24 альфа CDR3
<400> 600
Ala Met Arg Glu Gly Arg Gly Asp Ser Trp Gly Lys Leu Gln
1 5 10
<210> 601
<211> 293
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 24 - бета нативный
<400> 601
Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys Arg Thr Gly
1 5 10 15
Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His Glu Asn Met
20 25 30
Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu Ile Tyr Phe
35 40 45
Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro Glu Gly Tyr
50 55 60
Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile Leu Glu Ser
65 70 75 80
Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser Ser Pro Phe
85 90 95
Gly Arg Gly Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr
100 105 110
Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe
115 120 125
Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val
130 135 140
Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp
145 150 155 160
Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro
165 170 175
Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser
180 185 190
Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe
195 200 205
Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr
210 215 220
Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp
225 230 235 240
Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val
245 250 255
Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu
260 265 270
Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg
275 280 285
Lys Asp Ser Arg Gly
290
<210> 602
<211> 293
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 24 - бета, цистеин-модифицированный
<400> 602
Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys Arg Thr Gly
1 5 10 15
Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His Glu Asn Met
20 25 30
Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu Ile Tyr Phe
35 40 45
Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro Glu Gly Tyr
50 55 60
Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile Leu Glu Ser
65 70 75 80
Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser Ser Pro Phe
85 90 95
Gly Arg Gly Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr
100 105 110
Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe
115 120 125
Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val
130 135 140
Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp
145 150 155 160
Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro
165 170 175
Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser
180 185 190
Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe
195 200 205
Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr
210 215 220
Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp
225 230 235 240
Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val
245 250 255
Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu
260 265 270
Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg
275 280 285
Lys Asp Ser Arg Gly
290
<210> 603
<211> 312
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 24 - бета нативный
<400> 603
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Ser Pro Phe Gly Arg Gly Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr
115 120 125
Arg Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 604
<211> 312
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 24 - бета
цистеин-модифицированный
<400> 604
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Ser Pro Phe Gly Arg Gly Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr
115 120 125
Arg Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 605
<211> 114
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 24 бета, вариабельная область
<400> 605
Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys Arg Thr Gly
1 5 10 15
Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His Glu Asn Met
20 25 30
Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu Ile Tyr Phe
35 40 45
Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro Glu Gly Tyr
50 55 60
Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile Leu Glu Ser
65 70 75 80
Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser Ser Pro Phe
85 90 95
Gly Arg Gly Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr
100 105 110
Val Thr
<210> 606
<211> 13
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 24 бета CDR3
<400> 606
Ala Ser Ser Pro Phe Gly Arg Gly Ser Tyr Glu Gln Tyr
1 5 10
<210> 607
<211> 259
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 25 - альфа нативный
<400> 607
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
1 5 10 15
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln Ser Tyr
20 25 30
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
35 40 45
Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly Arg Tyr
50 55 60
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
65 70 75 80
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Arg Glu
85 90 95
Gly Ser Leu Thr Gly Gly Gly Asn Lys Leu Thr Phe Gly Thr Gly Thr
100 105 110
Gln Leu Lys Val Glu Leu Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr
115 120 125
Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr
130 135 140
Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val
145 150 155 160
Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys
165 170 175
Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala
180 185 190
Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser
195 200 205
Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr
210 215 220
Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile
225 230 235 240
Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu
245 250 255
Trp Ser Ser
<210> 608
<211> 259
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 25 - альфа, цистеин-модифицированный
<400> 608
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
1 5 10 15
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln Ser Tyr
20 25 30
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
35 40 45
Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly Arg Tyr
50 55 60
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
65 70 75 80
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Arg Glu
85 90 95
Gly Ser Leu Thr Gly Gly Gly Asn Lys Leu Thr Phe Gly Thr Gly Thr
100 105 110
Gln Leu Lys Val Glu Leu Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr
115 120 125
Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr
130 135 140
Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val
145 150 155 160
Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys
165 170 175
Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala
180 185 190
Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser
195 200 205
Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr
210 215 220
Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile
225 230 235 240
Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu
245 250 255
Trp Ser Ser
<210> 609
<211> 259
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 25 - альфа, цистеин-модифицированный
<400> 609
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
1 5 10 15
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln Ser Tyr
20 25 30
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
35 40 45
Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly Arg Tyr
50 55 60
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
65 70 75 80
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Arg Glu
85 90 95
Gly Ser Leu Thr Gly Gly Gly Asn Lys Leu Thr Phe Gly Thr Gly Thr
100 105 110
Gln Leu Lys Val Glu Leu Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr
115 120 125
Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr
130 135 140
Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val
145 150 155 160
Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys
165 170 175
Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala
180 185 190
Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser
195 200 205
Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr
210 215 220
Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile
225 230 235 240
Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu
245 250 255
Trp Ser Ser
<210> 610
<211> 279
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 25 - альфа, цистеин-модифицированный
<400> 610
Met Ser Leu Ser Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu
1 5 10 15
Gly Pro Gly Ile Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe
20 25 30
Val Gln Glu Lys Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser
35 40 45
Asp Gln Ser Tyr Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu
50 55 60
Met Ile Phe Leu Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr
65 70 75 80
Glu Gly Arg Tyr Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn
85 90 95
Leu Val Ile Ser Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys
100 105 110
Ala Met Arg Glu Gly Ser Leu Thr Gly Gly Gly Asn Lys Leu Thr Phe
115 120 125
Gly Thr Gly Thr Gln Leu Lys Val Glu Leu Asn Ile Gln Asn Pro Asp
130 135 140
Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val
145 150 155 160
Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys
165 170 175
Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser
180 185 190
Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp
195 200 205
Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr
210 215 220
Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys
225 230 235 240
Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile
245 250 255
Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met
260 265 270
Thr Leu Arg Leu Trp Ser Ser
275
<210> 611
<211> 118
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 25 альфа, вариабельная область
<400> 611
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
1 5 10 15
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln Ser Tyr
20 25 30
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
35 40 45
Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly Arg Tyr
50 55 60
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
65 70 75 80
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Arg Glu
85 90 95
Gly Ser Leu Thr Gly Gly Gly Asn Lys Leu Thr Phe Gly Thr Gly Thr
100 105 110
Gln Leu Lys Val Glu Leu
115
<210> 612
<211> 15
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 25 альфа CDR3
<400> 612
Ala Met Arg Glu Gly Ser Leu Thr Gly Gly Gly Asn Lys Leu Thr
1 5 10 15
<210> 613
<211> 291
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 25 - бета нативный
<400> 613
Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys Arg Thr Gly
1 5 10 15
Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His Glu Asn Met
20 25 30
Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu Ile Tyr Phe
35 40 45
Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro Glu Gly Tyr
50 55 60
Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile Leu Glu Ser
65 70 75 80
Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser Ser Leu Trp
85 90 95
Gly Gln Ser Asn Gln Pro Gln His Phe Gly Asp Gly Thr Arg Leu Ser
100 105 110
Ile Leu Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val Phe
115 120 125
Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val
130 135 140
Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp Trp
145 150 155 160
Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro
165 170 175
Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser
180 185 190
Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe
195 200 205
Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr
210 215 220
Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp
225 230 235 240
Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly Val
245 250 255
Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu
260 265 270
Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg
275 280 285
Lys Asp Phe
290
<210> 614
<211> 291
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 25 - бета, цистеин-модифицированный
<400> 614
Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys Arg Thr Gly
1 5 10 15
Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His Glu Asn Met
20 25 30
Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu Ile Tyr Phe
35 40 45
Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro Glu Gly Tyr
50 55 60
Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile Leu Glu Ser
65 70 75 80
Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser Ser Leu Trp
85 90 95
Gly Gln Ser Asn Gln Pro Gln His Phe Gly Asp Gly Thr Arg Leu Ser
100 105 110
Ile Leu Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val Phe
115 120 125
Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val
130 135 140
Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp Trp
145 150 155 160
Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro
165 170 175
Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser
180 185 190
Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe
195 200 205
Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr
210 215 220
Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp
225 230 235 240
Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly Val
245 250 255
Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu
260 265 270
Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg
275 280 285
Lys Asp Phe
290
<210> 615
<211> 310
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 25 - бета нативный
<400> 615
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Ser Leu Trp Gly Gln Ser Asn Gln Pro Gln His Phe Gly Asp Gly Thr
115 120 125
Arg Leu Ser Ile Leu Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe
305 310
<210> 616
<211> 310
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 25 - бета, цистеин-модифицированный
<400> 616
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Ser Leu Trp Gly Gln Ser Asn Gln Pro Gln His Phe Gly Asp Gly Thr
115 120 125
Arg Leu Ser Ile Leu Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe
305 310
<210> 617
<211> 114
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 25 бета, вариабельная область
<400> 617
Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys Arg Thr Gly
1 5 10 15
Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His Glu Asn Met
20 25 30
Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu Ile Tyr Phe
35 40 45
Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro Glu Gly Tyr
50 55 60
Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile Leu Glu Ser
65 70 75 80
Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser Ser Leu Trp
85 90 95
Gly Gln Ser Asn Gln Pro Gln His Phe Gly Asp Gly Thr Arg Leu Ser
100 105 110
Ile Leu
<210> 618
<211> 13
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 25 бета CDR3
<400> 618
Ala Ser Ser Leu Trp Gly Gln Ser Asn Gln Pro Gln His
1 5 10
<210> 619
<211> 255
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 26 - альфа нативный
<400> 619
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
1 5 10 15
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln Ser Tyr
20 25 30
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
35 40 45
Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly Arg Tyr
50 55 60
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
65 70 75 80
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Arg Asp
85 90 95
Ala Arg Asn Asn Asp Met Arg Phe Gly Ala Gly Thr Arg Leu Thr Val
100 105 110
Lys Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp
115 120 125
Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser
130 135 140
Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp
145 150 155 160
Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala
165 170 175
Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn
180 185 190
Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser
195 200 205
Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu
210 215 220
Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys
225 230 235 240
Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250 255
<210> 620
<211> 255
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 26 - альфа, цистеин-модифицированный
<400> 620
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
1 5 10 15
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln Ser Tyr
20 25 30
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
35 40 45
Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly Arg Tyr
50 55 60
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
65 70 75 80
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Arg Asp
85 90 95
Ala Arg Asn Asn Asp Met Arg Phe Gly Ala Gly Thr Arg Leu Thr Val
100 105 110
Lys Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp
115 120 125
Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser
130 135 140
Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp
145 150 155 160
Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala
165 170 175
Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn
180 185 190
Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser
195 200 205
Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu
210 215 220
Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys
225 230 235 240
Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250 255
<210> 621
<211> 275
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 26 - альфа нативный
<400> 621
Met Ser Leu Ser Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu
1 5 10 15
Gly Pro Gly Ile Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe
20 25 30
Val Gln Glu Lys Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser
35 40 45
Asp Gln Ser Tyr Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu
50 55 60
Met Ile Phe Leu Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr
65 70 75 80
Glu Gly Arg Tyr Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn
85 90 95
Leu Val Ile Ser Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys
100 105 110
Ala Met Arg Asp Ala Arg Asn Asn Asp Met Arg Phe Gly Ala Gly Thr
115 120 125
Arg Leu Thr Val Lys Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr
130 135 140
Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr
145 150 155 160
Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val
165 170 175
Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys
180 185 190
Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala
195 200 205
Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser
210 215 220
Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr
225 230 235 240
Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile
245 250 255
Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu
260 265 270
Trp Ser Ser
275
<210> 622
<211> 275
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 26 - альфа, цистеин-модифицированный
<400> 622
Met Ser Leu Ser Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu
1 5 10 15
Gly Pro Gly Ile Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe
20 25 30
Val Gln Glu Lys Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser
35 40 45
Asp Gln Ser Tyr Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu
50 55 60
Met Ile Phe Leu Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr
65 70 75 80
Glu Gly Arg Tyr Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn
85 90 95
Leu Val Ile Ser Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys
100 105 110
Ala Met Arg Asp Ala Arg Asn Asn Asp Met Arg Phe Gly Ala Gly Thr
115 120 125
Arg Leu Thr Val Lys Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr
130 135 140
Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr
145 150 155 160
Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val
165 170 175
Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys
180 185 190
Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala
195 200 205
Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser
210 215 220
Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr
225 230 235 240
Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile
245 250 255
Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu
260 265 270
Trp Ser Ser
275
<210> 623
<211> 114
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 26 альфа, вариабельная область
<400> 623
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
1 5 10 15
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln Ser Tyr
20 25 30
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
35 40 45
Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly Arg Tyr
50 55 60
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
65 70 75 80
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Arg Asp
85 90 95
Ala Arg Asn Asn Asp Met Arg Phe Gly Ala Gly Thr Arg Leu Thr Val
100 105 110
Lys Pro
<210> 624
<211> 11
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 26 альфа CDR3
<400> 624
Ala Met Arg Asp Ala Arg Asn Asn Asp Met Arg
1 5 10
<210> 625
<211> 294
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 26 - бета нативный
<400> 625
Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Arg Val Leu Lys Thr Gly
1 5 10 15
Gln Ser Met Thr Leu Leu Cys Ala Gln Asp Met Asn His Glu Tyr Met
20 25 30
Tyr Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu Ile His Tyr
35 40 45
Ser Val Gly Glu Gly Thr Thr Ala Lys Gly Glu Val Pro Asp Gly Tyr
50 55 60
Asn Val Ser Arg Leu Lys Lys Gln Asn Phe Leu Leu Gly Leu Glu Ser
65 70 75 80
Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser Ser Tyr Tyr
85 90 95
Ala Ser Gly Arg Asn Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu
100 105 110
Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val
115 120 125
Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu
130 135 140
Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp
145 150 155 160
Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln
165 170 175
Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser
180 185 190
Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His
195 200 205
Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp
210 215 220
Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala
225 230 235 240
Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly
245 250 255
Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr
260 265 270
Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys
275 280 285
Arg Lys Asp Ser Arg Gly
290
<210> 626
<211> 294
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 26 - бета, цистеин-модифицированный
<400> 626
Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Arg Val Leu Lys Thr Gly
1 5 10 15
Gln Ser Met Thr Leu Leu Cys Ala Gln Asp Met Asn His Glu Tyr Met
20 25 30
Tyr Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu Ile His Tyr
35 40 45
Ser Val Gly Glu Gly Thr Thr Ala Lys Gly Glu Val Pro Asp Gly Tyr
50 55 60
Asn Val Ser Arg Leu Lys Lys Gln Asn Phe Leu Leu Gly Leu Glu Ser
65 70 75 80
Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser Ser Tyr Tyr
85 90 95
Ala Ser Gly Arg Asn Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu
100 105 110
Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val
115 120 125
Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu
130 135 140
Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp
145 150 155 160
Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln
165 170 175
Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser
180 185 190
Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His
195 200 205
Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp
210 215 220
Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala
225 230 235 240
Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly
245 250 255
Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr
260 265 270
Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys
275 280 285
Arg Lys Asp Ser Arg Gly
290
<210> 627
<211> 313
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 26 - бета нативный
<400> 627
Met Ser Leu Gly Leu Leu Cys Cys Gly Ala Phe Ser Leu Leu Trp Ala
1 5 10 15
Gly Pro Val Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Arg Val Leu
20 25 30
Lys Thr Gly Gln Ser Met Thr Leu Leu Cys Ala Gln Asp Met Asn His
35 40 45
Glu Tyr Met Tyr Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu
50 55 60
Ile His Tyr Ser Val Gly Glu Gly Thr Thr Ala Lys Gly Glu Val Pro
65 70 75 80
Asp Gly Tyr Asn Val Ser Arg Leu Lys Lys Gln Asn Phe Leu Leu Gly
85 90 95
Leu Glu Ser Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser
100 105 110
Ser Tyr Tyr Ala Ser Gly Arg Asn Tyr Glu Gln Tyr Phe Gly Pro Gly
115 120 125
Thr Arg Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu
130 135 140
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
145 150 155 160
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu
165 170 175
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr
180 185 190
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
195 200 205
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
210 215 220
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
225 230 235 240
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
245 250 255
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr
260 265 270
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
275 280 285
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
290 295 300
Met Val Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 628
<211> 313
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 26 - бета, цистеин-модифицированный
<400> 628
Met Ser Leu Gly Leu Leu Cys Cys Gly Ala Phe Ser Leu Leu Trp Ala
1 5 10 15
Gly Pro Val Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Arg Val Leu
20 25 30
Lys Thr Gly Gln Ser Met Thr Leu Leu Cys Ala Gln Asp Met Asn His
35 40 45
Glu Tyr Met Tyr Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu
50 55 60
Ile His Tyr Ser Val Gly Glu Gly Thr Thr Ala Lys Gly Glu Val Pro
65 70 75 80
Asp Gly Tyr Asn Val Ser Arg Leu Lys Lys Gln Asn Phe Leu Leu Gly
85 90 95
Leu Glu Ser Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser
100 105 110
Ser Tyr Tyr Ala Ser Gly Arg Asn Tyr Glu Gln Tyr Phe Gly Pro Gly
115 120 125
Thr Arg Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu
130 135 140
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
145 150 155 160
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu
165 170 175
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr
180 185 190
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
195 200 205
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
210 215 220
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
225 230 235 240
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
245 250 255
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr
260 265 270
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
275 280 285
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
290 295 300
Met Val Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 629
<211> 114
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 26 бета, вариабельная область
<400> 629
Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Arg Val Leu Lys Thr Gly
1 5 10 15
Gln Ser Met Thr Leu Leu Cys Ala Gln Asp Met Asn His Glu Tyr Met
20 25 30
Tyr Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu Ile His Tyr
35 40 45
Ser Val Gly Glu Gly Thr Thr Ala Lys Gly Glu Val Pro Asp Gly Tyr
50 55 60
Asn Val Ser Arg Leu Lys Lys Gln Asn Phe Leu Leu Gly Leu Glu Ser
65 70 75 80
Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser Ser Tyr Tyr
85 90 95
Ala Ser Gly Arg Asn Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu
100 105 110
Thr Val
<210> 630
<211> 14
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 26 бета CDR3
<400> 630
Ala Ser Ser Tyr Tyr Ala Ser Gly Arg Asn Tyr Glu Gln Tyr
1 5 10
<210> 631
<211> 180
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 26 - нативный TCR бета, Константная область
<400> 631
Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu
1 5 10 15
Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys
20 25 30
Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val
35 40 45
Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro Leu
50 55 60
Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg
65 70 75 80
Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg
85 90 95
Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln
100 105 110
Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly
115 120 125
Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu
130 135 140
Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr
145 150 155 160
Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys
165 170 175
Asp Ser Arg Gly
180
<210> 632
<211> 180
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 26 - TCR бета, Константная область
<400> 632
Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu
1 5 10 15
Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys
20 25 30
Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val
35 40 45
Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro Leu
50 55 60
Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg
65 70 75 80
Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg
85 90 95
Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln
100 105 110
Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly
115 120 125
Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu
130 135 140
Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr
145 150 155 160
Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys
165 170 175
Asp Ser Arg Gly
180
<210> 633
<211> 255
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 27 - альфа нативный
<400> 633
Asp Ala Lys Thr Thr Gln Pro Asn Ser Met Glu Ser Asn Glu Glu Glu
1 5 10 15
Pro Val His Leu Pro Cys Asn His Ser Thr Ile Ser Gly Thr Asp Tyr
20 25 30
Ile His Trp Tyr Arg Gln Leu Pro Ser Gln Gly Pro Glu Tyr Val Ile
35 40 45
His Gly Leu Thr Ser Asn Val Asn Asn Arg Met Ala Ser Leu Ala Ile
50 55 60
Ala Glu Asp Arg Lys Ser Ser Thr Leu Ile Leu His Arg Ala Thr Leu
65 70 75 80
Arg Asp Ala Ala Val Tyr Tyr Cys Ile Leu Arg Val Pro Pro Gln Ser
85 90 95
Gly Gly Tyr Gln Lys Val Thr Phe Gly Thr Gly Thr Lys Leu Gln Val
100 105 110
Ile Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp
115 120 125
Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser
130 135 140
Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp
145 150 155 160
Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala
165 170 175
Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn
180 185 190
Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser
195 200 205
Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu
210 215 220
Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys
225 230 235 240
Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250 255
<210> 634
<211> 255
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 27 - альфа, цистеин-модифицированный
<400> 634
Asp Ala Lys Thr Thr Gln Pro Asn Ser Met Glu Ser Asn Glu Glu Glu
1 5 10 15
Pro Val His Leu Pro Cys Asn His Ser Thr Ile Ser Gly Thr Asp Tyr
20 25 30
Ile His Trp Tyr Arg Gln Leu Pro Ser Gln Gly Pro Glu Tyr Val Ile
35 40 45
His Gly Leu Thr Ser Asn Val Asn Asn Arg Met Ala Ser Leu Ala Ile
50 55 60
Ala Glu Asp Arg Lys Ser Ser Thr Leu Ile Leu His Arg Ala Thr Leu
65 70 75 80
Arg Asp Ala Ala Val Tyr Tyr Cys Ile Leu Arg Val Pro Pro Gln Ser
85 90 95
Gly Gly Tyr Gln Lys Val Thr Phe Gly Thr Gly Thr Lys Leu Gln Val
100 105 110
Ile Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp
115 120 125
Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser
130 135 140
Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp
145 150 155 160
Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala
165 170 175
Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn
180 185 190
Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser
195 200 205
Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu
210 215 220
Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys
225 230 235 240
Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250 255
<210> 635
<211> 272
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 27 - альфа нативный
<400> 635
Met Lys Leu Val Thr Ser Ile Thr Val Leu Leu Ser Leu Gly Ile Met
1 5 10 15
Gly Asp Ala Lys Thr Thr Gln Pro Asn Ser Met Glu Ser Asn Glu Glu
20 25 30
Glu Pro Val His Leu Pro Cys Asn His Ser Thr Ile Ser Gly Thr Asp
35 40 45
Tyr Ile His Trp Tyr Arg Gln Leu Pro Ser Gln Gly Pro Glu Tyr Val
50 55 60
Ile His Gly Leu Thr Ser Asn Val Asn Asn Arg Met Ala Ser Leu Ala
65 70 75 80
Ile Ala Glu Asp Arg Lys Ser Ser Thr Leu Ile Leu His Arg Ala Thr
85 90 95
Leu Arg Asp Ala Ala Val Tyr Tyr Cys Ile Leu Arg Val Pro Pro Gln
100 105 110
Ser Gly Gly Tyr Gln Lys Val Thr Phe Gly Thr Gly Thr Lys Leu Gln
115 120 125
Val Ile Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg
130 135 140
Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp
145 150 155 160
Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr
165 170 175
Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser
180 185 190
Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe
195 200 205
Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser
210 215 220
Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn
225 230 235 240
Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu
245 250 255
Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
260 265 270
<210> 636
<211> 272
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 27 - альфа, цистеин-модифицированный
<400> 636
Met Lys Leu Val Thr Ser Ile Thr Val Leu Leu Ser Leu Gly Ile Met
1 5 10 15
Gly Asp Ala Lys Thr Thr Gln Pro Asn Ser Met Glu Ser Asn Glu Glu
20 25 30
Glu Pro Val His Leu Pro Cys Asn His Ser Thr Ile Ser Gly Thr Asp
35 40 45
Tyr Ile His Trp Tyr Arg Gln Leu Pro Ser Gln Gly Pro Glu Tyr Val
50 55 60
Ile His Gly Leu Thr Ser Asn Val Asn Asn Arg Met Ala Ser Leu Ala
65 70 75 80
Ile Ala Glu Asp Arg Lys Ser Ser Thr Leu Ile Leu His Arg Ala Thr
85 90 95
Leu Arg Asp Ala Ala Val Tyr Tyr Cys Ile Leu Arg Val Pro Pro Gln
100 105 110
Ser Gly Gly Tyr Gln Lys Val Thr Phe Gly Thr Gly Thr Lys Leu Gln
115 120 125
Val Ile Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg
130 135 140
Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp
145 150 155 160
Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr
165 170 175
Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser
180 185 190
Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe
195 200 205
Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser
210 215 220
Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn
225 230 235 240
Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu
245 250 255
Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
260 265 270
<210> 637
<211> 114
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 27 альфа, вариабельная область
<400> 637
Asp Ala Lys Thr Thr Gln Pro Asn Ser Met Glu Ser Asn Glu Glu Glu
1 5 10 15
Pro Val His Leu Pro Cys Asn His Ser Thr Ile Ser Gly Thr Asp Tyr
20 25 30
Ile His Trp Tyr Arg Gln Leu Pro Ser Gln Gly Pro Glu Tyr Val Ile
35 40 45
His Gly Leu Thr Ser Asn Val Asn Asn Arg Met Ala Ser Leu Ala Ile
50 55 60
Ala Glu Asp Arg Lys Ser Ser Thr Leu Ile Leu His Arg Ala Thr Leu
65 70 75 80
Arg Asp Ala Ala Val Tyr Tyr Cys Ile Leu Arg Val Pro Pro Gln Ser
85 90 95
Gly Gly Tyr Gln Lys Val Thr Phe Gly Thr Gly Thr Lys Leu Gln Val
100 105 110
Ile Pro
<210> 638
<211> 15
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 27 альфа CDR3
<400> 638
Ile Leu Arg Val Pro Pro Gln Ser Gly Gly Tyr Gln Lys Val Thr
1 5 10 15
<210> 639
<211> 291
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 27 - бета нативный
<400> 639
Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Gln Val Leu Lys Thr Gly
1 5 10 15
Gln Ser Met Thr Leu Gln Cys Ala Gln Asp Met Asn His Asn Ser Met
20 25 30
Tyr Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu Ile Tyr Tyr
35 40 45
Ser Ala Ser Glu Gly Thr Thr Asp Lys Gly Glu Val Pro Asn Gly Tyr
50 55 60
Asn Val Ser Arg Leu Asn Lys Arg Glu Phe Ser Leu Arg Leu Glu Ser
65 70 75 80
Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser Ser Glu Phe
85 90 95
Gly Ser Leu Asn Glu Lys Leu Phe Phe Gly Ser Gly Thr Gln Leu Ser
100 105 110
Val Leu Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val Phe
115 120 125
Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val
130 135 140
Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp Trp
145 150 155 160
Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro
165 170 175
Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser
180 185 190
Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe
195 200 205
Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr
210 215 220
Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp
225 230 235 240
Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly Val
245 250 255
Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu
260 265 270
Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg
275 280 285
Lys Asp Phe
290
<210> 640
<211> 291
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 27 - бета, цистеин-модифицированный
<400> 640
Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Gln Val Leu Lys Thr Gly
1 5 10 15
Gln Ser Met Thr Leu Gln Cys Ala Gln Asp Met Asn His Asn Ser Met
20 25 30
Tyr Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu Ile Tyr Tyr
35 40 45
Ser Ala Ser Glu Gly Thr Thr Asp Lys Gly Glu Val Pro Asn Gly Tyr
50 55 60
Asn Val Ser Arg Leu Asn Lys Arg Glu Phe Ser Leu Arg Leu Glu Ser
65 70 75 80
Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser Ser Glu Phe
85 90 95
Gly Ser Leu Asn Glu Lys Leu Phe Phe Gly Ser Gly Thr Gln Leu Ser
100 105 110
Val Leu Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val Phe
115 120 125
Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val
130 135 140
Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp Trp
145 150 155 160
Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro
165 170 175
Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser
180 185 190
Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe
195 200 205
Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr
210 215 220
Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp
225 230 235 240
Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly Val
245 250 255
Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu
260 265 270
Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg
275 280 285
Lys Asp Phe
290
<210> 641
<211> 324
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 27 - бета нативный
<400> 641
Met His Arg Pro Arg Arg Pro Leu His Pro Val Ala Pro Ala Met Ser
1 5 10 15
Ile Gly Leu Leu Cys Cys Val Ala Phe Ser Leu Leu Trp Ala Ser Pro
20 25 30
Val Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Gln Val Leu Lys Thr
35 40 45
Gly Gln Ser Met Thr Leu Gln Cys Ala Gln Asp Met Asn His Asn Ser
50 55 60
Met Tyr Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu Ile Tyr
65 70 75 80
Tyr Ser Ala Ser Glu Gly Thr Thr Asp Lys Gly Glu Val Pro Asn Gly
85 90 95
Tyr Asn Val Ser Arg Leu Asn Lys Arg Glu Phe Ser Leu Arg Leu Glu
100 105 110
Ser Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser Ser Glu
115 120 125
Phe Gly Ser Leu Asn Glu Lys Leu Phe Phe Gly Ser Gly Thr Gln Leu
130 135 140
Ser Val Leu Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val
145 150 155 160
Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu
165 170 175
Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp
180 185 190
Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln
195 200 205
Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser
210 215 220
Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His
225 230 235 240
Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp
245 250 255
Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala
260 265 270
Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly
275 280 285
Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr
290 295 300
Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys
305 310 315 320
Arg Lys Asp Phe
<210> 642
<211> 324
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 27 - бета, цистеин-модифицированный
<400> 642
Met His Arg Pro Arg Arg Pro Leu His Pro Val Ala Pro Ala Met Ser
1 5 10 15
Ile Gly Leu Leu Cys Cys Val Ala Phe Ser Leu Leu Trp Ala Ser Pro
20 25 30
Val Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Gln Val Leu Lys Thr
35 40 45
Gly Gln Ser Met Thr Leu Gln Cys Ala Gln Asp Met Asn His Asn Ser
50 55 60
Met Tyr Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu Ile Tyr
65 70 75 80
Tyr Ser Ala Ser Glu Gly Thr Thr Asp Lys Gly Glu Val Pro Asn Gly
85 90 95
Tyr Asn Val Ser Arg Leu Asn Lys Arg Glu Phe Ser Leu Arg Leu Glu
100 105 110
Ser Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser Ser Glu
115 120 125
Phe Gly Ser Leu Asn Glu Lys Leu Phe Phe Gly Ser Gly Thr Gln Leu
130 135 140
Ser Val Leu Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val
145 150 155 160
Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu
165 170 175
Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp
180 185 190
Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln
195 200 205
Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser
210 215 220
Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His
225 230 235 240
Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp
245 250 255
Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala
260 265 270
Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly
275 280 285
Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr
290 295 300
Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys
305 310 315 320
Arg Lys Asp Phe
<210> 643
<211> 114
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 27 бета, вариабельная область
<400> 643
Asn Ala Gly Val Thr Gln Thr Pro Lys Phe Gln Val Leu Lys Thr Gly
1 5 10 15
Gln Ser Met Thr Leu Gln Cys Ala Gln Asp Met Asn His Asn Ser Met
20 25 30
Tyr Trp Tyr Arg Gln Asp Pro Gly Met Gly Leu Arg Leu Ile Tyr Tyr
35 40 45
Ser Ala Ser Glu Gly Thr Thr Asp Lys Gly Glu Val Pro Asn Gly Tyr
50 55 60
Asn Val Ser Arg Leu Asn Lys Arg Glu Phe Ser Leu Arg Leu Glu Ser
65 70 75 80
Ala Ala Pro Ser Gln Thr Ser Val Tyr Phe Cys Ala Ser Ser Glu Phe
85 90 95
Gly Ser Leu Asn Glu Lys Leu Phe Phe Gly Ser Gly Thr Gln Leu Ser
100 105 110
Val Leu
<210> 644
<211> 13
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 27 бета CDR3
<400> 644
Ala Ser Ser Glu Phe Gly Ser Leu Asn Glu Lys Leu Phe
1 5 10
<210> 645
<211> 253
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 28 - альфа нативный
<400> 645
Arg Lys Glu Val Glu Gln Asp Pro Gly Pro Phe Asn Val Pro Glu Gly
1 5 10 15
Ala Thr Val Ala Phe Asn Cys Thr Tyr Ser Asn Ser Ala Ser Gln Ser
20 25 30
Phe Phe Trp Tyr Arg Gln Asp Cys Arg Lys Glu Pro Lys Leu Leu Met
35 40 45
Ser Val Tyr Ser Ser Gly Asn Glu Asp Gly Arg Phe Thr Ala Gln Leu
50 55 60
Asn Arg Ala Ser Gln Tyr Ile Ser Leu Leu Ile Arg Asp Ser Lys Leu
65 70 75 80
Ser Asp Ser Ala Thr Tyr Leu Cys Val Val Arg Gly Gly Gly Thr Ser
85 90 95
Tyr Gly Lys Leu Thr Phe Gly Gln Gly Thr Ile Leu Thr Val His Pro
100 105 110
Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys
115 120 125
Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr
130 135 140
Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr
145 150 155 160
Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala
165 170 175
Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser
180 185 190
Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp
195 200 205
Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe
210 215 220
Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala
225 230 235 240
Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 646
<211> 253
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 28 - альфа, цистеин-модифицированный
<400> 646
Arg Lys Glu Val Glu Gln Asp Pro Gly Pro Phe Asn Val Pro Glu Gly
1 5 10 15
Ala Thr Val Ala Phe Asn Cys Thr Tyr Ser Asn Ser Ala Ser Gln Ser
20 25 30
Phe Phe Trp Tyr Arg Gln Asp Cys Arg Lys Glu Pro Lys Leu Leu Met
35 40 45
Ser Val Tyr Ser Ser Gly Asn Glu Asp Gly Arg Phe Thr Ala Gln Leu
50 55 60
Asn Arg Ala Ser Gln Tyr Ile Ser Leu Leu Ile Arg Asp Ser Lys Leu
65 70 75 80
Ser Asp Ser Ala Thr Tyr Leu Cys Val Val Arg Gly Gly Gly Thr Ser
85 90 95
Tyr Gly Lys Leu Thr Phe Gly Gln Gly Thr Ile Leu Thr Val His Pro
100 105 110
Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys
115 120 125
Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr
130 135 140
Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys
145 150 155 160
Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala
165 170 175
Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser
180 185 190
Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp
195 200 205
Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe
210 215 220
Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala
225 230 235 240
Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 647
<211> 275
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 28 - альфа нативный
<400> 647
Met Met Ile Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu
1 5 10 15
Ser Trp Val Trp Ser Gln Arg Lys Glu Val Glu Gln Asp Pro Gly Pro
20 25 30
Phe Asn Val Pro Glu Gly Ala Thr Val Ala Phe Asn Cys Thr Tyr Ser
35 40 45
Asn Ser Ala Ser Gln Ser Phe Phe Trp Tyr Arg Gln Asp Cys Arg Lys
50 55 60
Glu Pro Lys Leu Leu Met Ser Val Tyr Ser Ser Gly Asn Glu Asp Gly
65 70 75 80
Arg Phe Thr Ala Gln Leu Asn Arg Ala Ser Gln Tyr Ile Ser Leu Leu
85 90 95
Ile Arg Asp Ser Lys Leu Ser Asp Ser Ala Thr Tyr Leu Cys Val Val
100 105 110
Arg Gly Gly Gly Thr Ser Tyr Gly Lys Leu Thr Phe Gly Gln Gly Thr
115 120 125
Ile Leu Thr Val His Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr
130 135 140
Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr
145 150 155 160
Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val
165 170 175
Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys
180 185 190
Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala
195 200 205
Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser
210 215 220
Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr
225 230 235 240
Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile
245 250 255
Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu
260 265 270
Trp Ser Ser
275
<210> 648
<211> 275
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 28 - альфа, цистеин-модифицированный
<400> 648
Met Met Ile Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu
1 5 10 15
Ser Trp Val Trp Ser Gln Arg Lys Glu Val Glu Gln Asp Pro Gly Pro
20 25 30
Phe Asn Val Pro Glu Gly Ala Thr Val Ala Phe Asn Cys Thr Tyr Ser
35 40 45
Asn Ser Ala Ser Gln Ser Phe Phe Trp Tyr Arg Gln Asp Cys Arg Lys
50 55 60
Glu Pro Lys Leu Leu Met Ser Val Tyr Ser Ser Gly Asn Glu Asp Gly
65 70 75 80
Arg Phe Thr Ala Gln Leu Asn Arg Ala Ser Gln Tyr Ile Ser Leu Leu
85 90 95
Ile Arg Asp Ser Lys Leu Ser Asp Ser Ala Thr Tyr Leu Cys Val Val
100 105 110
Arg Gly Gly Gly Thr Ser Tyr Gly Lys Leu Thr Phe Gly Gln Gly Thr
115 120 125
Ile Leu Thr Val His Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr
130 135 140
Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr
145 150 155 160
Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val
165 170 175
Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys
180 185 190
Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala
195 200 205
Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser
210 215 220
Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr
225 230 235 240
Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile
245 250 255
Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu
260 265 270
Trp Ser Ser
275
<210> 649
<211> 112
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 28 альфа, вариабельная область
<400> 649
Arg Lys Glu Val Glu Gln Asp Pro Gly Pro Phe Asn Val Pro Glu Gly
1 5 10 15
Ala Thr Val Ala Phe Asn Cys Thr Tyr Ser Asn Ser Ala Ser Gln Ser
20 25 30
Phe Phe Trp Tyr Arg Gln Asp Cys Arg Lys Glu Pro Lys Leu Leu Met
35 40 45
Ser Val Tyr Ser Ser Gly Asn Glu Asp Gly Arg Phe Thr Ala Gln Leu
50 55 60
Asn Arg Ala Ser Gln Tyr Ile Ser Leu Leu Ile Arg Asp Ser Lys Leu
65 70 75 80
Ser Asp Ser Ala Thr Tyr Leu Cys Val Val Arg Gly Gly Gly Thr Ser
85 90 95
Tyr Gly Lys Leu Thr Phe Gly Gln Gly Thr Ile Leu Thr Val His Pro
100 105 110
<210> 650
<211> 13
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 28 альфа CDR3
<400> 650
Val Val Arg Gly Gly Gly Thr Ser Tyr Gly Lys Leu Thr
1 5 10
<210> 651
<211> 292
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 28 - бета нативный
<400> 651
Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys Arg Thr Gly
1 5 10 15
Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His Glu Asn Met
20 25 30
Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu Ile Tyr Phe
35 40 45
Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro Glu Gly Tyr
50 55 60
Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile Leu Glu Ser
65 70 75 80
Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser Ser Leu Trp
85 90 95
Gly Arg Ser Ser Gly Asn Thr Ile Tyr Phe Gly Glu Gly Ser Trp Leu
100 105 110
Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val
115 120 125
Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu
130 135 140
Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp
145 150 155 160
Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln
165 170 175
Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser
180 185 190
Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His
195 200 205
Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp
210 215 220
Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala
225 230 235 240
Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly
245 250 255
Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr
260 265 270
Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys
275 280 285
Arg Lys Asp Phe
290
<210> 652
<211> 292
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 28 - бета, цистеин-модифицированный
<400> 652
Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys Arg Thr Gly
1 5 10 15
Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His Glu Asn Met
20 25 30
Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu Ile Tyr Phe
35 40 45
Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro Glu Gly Tyr
50 55 60
Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile Leu Glu Ser
65 70 75 80
Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser Ser Leu Trp
85 90 95
Gly Arg Ser Ser Gly Asn Thr Ile Tyr Phe Gly Glu Gly Ser Trp Leu
100 105 110
Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val
115 120 125
Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu
130 135 140
Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp
145 150 155 160
Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln
165 170 175
Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser
180 185 190
Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His
195 200 205
Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp
210 215 220
Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala
225 230 235 240
Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly
245 250 255
Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr
260 265 270
Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys
275 280 285
Arg Lys Asp Phe
290
<210> 653
<211> 311
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 28 - бета нативный
<400> 653
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Ser Leu Trp Gly Arg Ser Ser Gly Asn Thr Ile Tyr Phe Gly Glu Gly
115 120 125
Ser Trp Leu Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu
130 135 140
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
145 150 155 160
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu
165 170 175
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr
180 185 190
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
195 200 205
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
210 215 220
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
225 230 235 240
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
245 250 255
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr
260 265 270
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
275 280 285
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
290 295 300
Met Val Lys Arg Lys Asp Phe
305 310
<210> 654
<211> 311
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 28 - бета, цистеин-модифицированный
<400> 654
Met Gly Ile Arg Leu Leu Cys Arg Val Ala Phe Cys Phe Leu Ala Val
1 5 10 15
Gly Leu Val Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys
20 25 30
Arg Thr Gly Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His
35 40 45
Glu Asn Met Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu
50 55 60
Ile Tyr Phe Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile
85 90 95
Leu Glu Ser Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser
100 105 110
Ser Leu Trp Gly Arg Ser Ser Gly Asn Thr Ile Tyr Phe Gly Glu Gly
115 120 125
Ser Trp Leu Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu
130 135 140
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
145 150 155 160
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu
165 170 175
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr
180 185 190
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
195 200 205
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
210 215 220
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
225 230 235 240
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
245 250 255
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr
260 265 270
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
275 280 285
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
290 295 300
Met Val Lys Arg Lys Asp Phe
305 310
<210> 655
<211> 115
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 28 бета, вариабельная область
<400> 655
Asp Val Lys Val Thr Gln Ser Ser Arg Tyr Leu Val Lys Arg Thr Gly
1 5 10 15
Glu Lys Val Phe Leu Glu Cys Val Gln Asp Met Asp His Glu Asn Met
20 25 30
Phe Trp Tyr Arg Gln Asp Pro Gly Leu Gly Leu Arg Leu Ile Tyr Phe
35 40 45
Ser Tyr Asp Val Lys Met Lys Glu Lys Gly Asp Ile Pro Glu Gly Tyr
50 55 60
Ser Val Ser Arg Glu Lys Lys Glu Arg Phe Ser Leu Ile Leu Glu Ser
65 70 75 80
Ala Ser Thr Asn Gln Thr Ser Met Tyr Leu Cys Ala Ser Ser Leu Trp
85 90 95
Gly Arg Ser Ser Gly Asn Thr Ile Tyr Phe Gly Glu Gly Ser Trp Leu
100 105 110
Thr Val Val
115
<210> 656
<211> 14
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 28 бета CDR3
<400> 656
Ala Ser Ser Leu Trp Gly Arg Ser Ser Gly Asn Thr Ile Tyr
1 5 10
<210> 657
<211> 257
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 29 - альфа нативный
<400> 657
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
1 5 10 15
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln Ser Tyr
20 25 30
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
35 40 45
Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly Arg Tyr
50 55 60
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
65 70 75 80
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Arg Leu
85 90 95
Asn Thr Gly Thr Ala Ser Lys Leu Thr Phe Gly Thr Gly Thr Arg Leu
100 105 110
Gln Val Thr Leu Asp Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu
115 120 125
Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe
130 135 140
Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile
145 150 155 160
Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn
165 170 175
Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala
180 185 190
Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu
195 200 205
Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr
210 215 220
Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu
225 230 235 240
Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser
245 250 255
Ser
<210> 658
<211> 257
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 29 - альфа, цистеин-модифицированный
<400> 658
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
1 5 10 15
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln Ser Tyr
20 25 30
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
35 40 45
Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly Arg Tyr
50 55 60
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
65 70 75 80
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Arg Leu
85 90 95
Asn Thr Gly Thr Ala Ser Lys Leu Thr Phe Gly Thr Gly Thr Arg Leu
100 105 110
Gln Val Thr Leu Asp Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu
115 120 125
Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe
130 135 140
Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile
145 150 155 160
Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn
165 170 175
Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala
180 185 190
Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu
195 200 205
Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr
210 215 220
Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu
225 230 235 240
Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser
245 250 255
Ser
<210> 659
<211> 277
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 29 - альфа нативный
<400> 659
Met Ser Leu Ser Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu
1 5 10 15
Gly Pro Gly Ile Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe
20 25 30
Val Gln Glu Lys Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser
35 40 45
Asp Gln Ser Tyr Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu
50 55 60
Met Ile Phe Leu Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr
65 70 75 80
Glu Gly Arg Tyr Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn
85 90 95
Leu Val Ile Ser Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys
100 105 110
Ala Met Arg Leu Asn Thr Gly Thr Ala Ser Lys Leu Thr Phe Gly Thr
115 120 125
Gly Thr Arg Leu Gln Val Thr Leu Asp Ile Gln Asn Pro Asp Pro Ala
130 135 140
Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu
145 150 155 160
Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser
165 170 175
Asp Val Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp
180 185 190
Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala
195 200 205
Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe
210 215 220
Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe
225 230 235 240
Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe
245 250 255
Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu
260 265 270
Arg Leu Trp Ser Ser
275
<210> 660
<211> 277
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 29 - альфа
цистеин-модифицированный
<400> 660
Met Ser Leu Ser Ser Leu Leu Lys Val Val Thr Ala Ser Leu Trp Leu
1 5 10 15
Gly Pro Gly Ile Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe
20 25 30
Val Gln Glu Lys Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser
35 40 45
Asp Gln Ser Tyr Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu
50 55 60
Met Ile Phe Leu Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr
65 70 75 80
Glu Gly Arg Tyr Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn
85 90 95
Leu Val Ile Ser Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys
100 105 110
Ala Met Arg Leu Asn Thr Gly Thr Ala Ser Lys Leu Thr Phe Gly Thr
115 120 125
Gly Thr Arg Leu Gln Val Thr Leu Asp Ile Gln Asn Pro Asp Pro Ala
130 135 140
Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu
145 150 155 160
Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser
165 170 175
Asp Val Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp
180 185 190
Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala
195 200 205
Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe
210 215 220
Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe
225 230 235 240
Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe
245 250 255
Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu
260 265 270
Arg Leu Trp Ser Ser
275
<210> 661
<211> 116
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 29 альфа, вариабельная область
<400> 661
Ala Gln Lys Ile Thr Gln Thr Gln Pro Gly Met Phe Val Gln Glu Lys
1 5 10 15
Glu Ala Val Thr Leu Asp Cys Thr Tyr Asp Thr Ser Asp Gln Ser Tyr
20 25 30
Gly Leu Phe Trp Tyr Lys Gln Pro Ser Ser Gly Glu Met Ile Phe Leu
35 40 45
Ile Tyr Gln Gly Ser Tyr Asp Glu Gln Asn Ala Thr Glu Gly Arg Tyr
50 55 60
Ser Leu Asn Phe Gln Lys Ala Arg Lys Ser Ala Asn Leu Val Ile Ser
65 70 75 80
Ala Ser Gln Leu Gly Asp Ser Ala Met Tyr Phe Cys Ala Met Arg Leu
85 90 95
Asn Thr Gly Thr Ala Ser Lys Leu Thr Phe Gly Thr Gly Thr Arg Leu
100 105 110
Gln Val Thr Leu
115
<210> 662
<211> 13
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 29 альфа CDR3
<400> 662
Ala Met Arg Leu Asn Thr Gly Thr Ala Ser Lys Leu Thr
1 5 10
<210> 663
<211> 293
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 29 - бета нативный
<400> 663
Asp Gly Gly Ile Thr Gln Ser Pro Lys Tyr Leu Phe Arg Lys Glu Gly
1 5 10 15
Gln Asn Val Thr Leu Ser Cys Glu Gln Asn Leu Asn His Asp Ala Met
20 25 30
Tyr Trp Tyr Arg Gln Asp Pro Gly Gln Gly Leu Arg Leu Ile Tyr Tyr
35 40 45
Ser Gln Ile Val Asn Asp Phe Gln Lys Gly Asp Ile Ala Glu Gly Tyr
50 55 60
Ser Val Ser Arg Glu Lys Lys Glu Ser Phe Pro Leu Thr Val Thr Ser
65 70 75 80
Ala Gln Lys Asn Pro Thr Ala Phe Tyr Leu Cys Ala Ser Ser Pro Trp
85 90 95
Gly Arg Ala Thr Asn Glu Gln Phe Phe Gly Pro Gly Thr Arg Leu Thr
100 105 110
Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe
115 120 125
Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val
130 135 140
Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp
145 150 155 160
Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro
165 170 175
Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser
180 185 190
Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe
195 200 205
Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr
210 215 220
Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp
225 230 235 240
Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val
245 250 255
Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu
260 265 270
Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg
275 280 285
Lys Asp Ser Arg Gly
290
<210> 664
<211> 293
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 29 - бета, цистеин-модифицированный
<400> 664
Asp Gly Gly Ile Thr Gln Ser Pro Lys Tyr Leu Phe Arg Lys Glu Gly
1 5 10 15
Gln Asn Val Thr Leu Ser Cys Glu Gln Asn Leu Asn His Asp Ala Met
20 25 30
Tyr Trp Tyr Arg Gln Asp Pro Gly Gln Gly Leu Arg Leu Ile Tyr Tyr
35 40 45
Ser Gln Ile Val Asn Asp Phe Gln Lys Gly Asp Ile Ala Glu Gly Tyr
50 55 60
Ser Val Ser Arg Glu Lys Lys Glu Ser Phe Pro Leu Thr Val Thr Ser
65 70 75 80
Ala Gln Lys Asn Pro Thr Ala Phe Tyr Leu Cys Ala Ser Ser Pro Trp
85 90 95
Gly Arg Ala Thr Asn Glu Gln Phe Phe Gly Pro Gly Thr Arg Leu Thr
100 105 110
Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe
115 120 125
Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val
130 135 140
Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp
145 150 155 160
Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro
165 170 175
Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser
180 185 190
Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe
195 200 205
Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr
210 215 220
Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp
225 230 235 240
Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val
245 250 255
Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu
260 265 270
Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg
275 280 285
Lys Asp Ser Arg Gly
290
<210> 665
<211> 312
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 29 - бета нативный
<400> 665
Met Ser Asn Gln Val Leu Cys Cys Val Val Leu Cys Leu Leu Gly Ala
1 5 10 15
Asn Thr Val Asp Gly Gly Ile Thr Gln Ser Pro Lys Tyr Leu Phe Arg
20 25 30
Lys Glu Gly Gln Asn Val Thr Leu Ser Cys Glu Gln Asn Leu Asn His
35 40 45
Asp Ala Met Tyr Trp Tyr Arg Gln Asp Pro Gly Gln Gly Leu Arg Leu
50 55 60
Ile Tyr Tyr Ser Gln Ile Val Asn Asp Phe Gln Lys Gly Asp Ile Ala
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Ser Phe Pro Leu Thr
85 90 95
Val Thr Ser Ala Gln Lys Asn Pro Thr Ala Phe Tyr Leu Cys Ala Ser
100 105 110
Ser Pro Trp Gly Arg Ala Thr Asn Glu Gln Phe Phe Gly Pro Gly Thr
115 120 125
Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 666
<211> 312
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 29 - бета, цистеин-модифицированный
<400> 666
Met Ser Asn Gln Val Leu Cys Cys Val Val Leu Cys Leu Leu Gly Ala
1 5 10 15
Asn Thr Val Asp Gly Gly Ile Thr Gln Ser Pro Lys Tyr Leu Phe Arg
20 25 30
Lys Glu Gly Gln Asn Val Thr Leu Ser Cys Glu Gln Asn Leu Asn His
35 40 45
Asp Ala Met Tyr Trp Tyr Arg Gln Asp Pro Gly Gln Gly Leu Arg Leu
50 55 60
Ile Tyr Tyr Ser Gln Ile Val Asn Asp Phe Gln Lys Gly Asp Ile Ala
65 70 75 80
Glu Gly Tyr Ser Val Ser Arg Glu Lys Lys Glu Ser Phe Pro Leu Thr
85 90 95
Val Thr Ser Ala Gln Lys Asn Pro Thr Ala Phe Tyr Leu Cys Ala Ser
100 105 110
Ser Pro Trp Gly Arg Ala Thr Asn Glu Gln Phe Phe Gly Pro Gly Thr
115 120 125
Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 667
<211> 114
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 29 бета, вариабельная область
<400> 667
Asp Gly Gly Ile Thr Gln Ser Pro Lys Tyr Leu Phe Arg Lys Glu Gly
1 5 10 15
Gln Asn Val Thr Leu Ser Cys Glu Gln Asn Leu Asn His Asp Ala Met
20 25 30
Tyr Trp Tyr Arg Gln Asp Pro Gly Gln Gly Leu Arg Leu Ile Tyr Tyr
35 40 45
Ser Gln Ile Val Asn Asp Phe Gln Lys Gly Asp Ile Ala Glu Gly Tyr
50 55 60
Ser Val Ser Arg Glu Lys Lys Glu Ser Phe Pro Leu Thr Val Thr Ser
65 70 75 80
Ala Gln Lys Asn Pro Thr Ala Phe Tyr Leu Cys Ala Ser Ser Pro Trp
85 90 95
Gly Arg Ala Thr Asn Glu Gln Phe Phe Gly Pro Gly Thr Arg Leu Thr
100 105 110
Val Leu
<210> 668
<211> 5
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 29 бета CDR1
<400> 668
Leu Asn His Asp Ala
1 5
<210> 669
<211> 6
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 29 бета CDR2
<400> 669
Ser Gln Ile Val Asn Asp
1 5
<210> 670
<211> 13
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 29 бета CDR3
<400> 670
Ala Ser Ser Pro Trp Gly Arg Ala Thr Asn Glu Gln Phe
1 5 10
<210> 671
<211> 19
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 29 бета, Сигнальный пептид
<400> 671
Met Ser Asn Gln Val Leu Cys Cys Val Val Leu Cys Leu Leu Gly Ala
1 5 10 15
Asn Thr Val
<210> 672
<211> 255
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 30 - альфа нативный
<400> 672
Asp Ala Lys Thr Thr Gln Pro Pro Ser Met Asp Cys Ala Glu Gly Arg
1 5 10 15
Ala Ala Asn Leu Pro Cys Asn His Ser Thr Ile Ser Gly Asn Glu Tyr
20 25 30
Val Tyr Trp Tyr Arg Gln Ile His Ser Gln Gly Pro Gln Tyr Ile Ile
35 40 45
His Gly Leu Lys Asn Asn Glu Thr Asn Glu Met Ala Ser Leu Ile Ile
50 55 60
Thr Glu Asp Arg Lys Ser Ser Thr Leu Ile Leu Pro His Ala Thr Leu
65 70 75 80
Arg Asp Thr Ala Val Tyr Tyr Cys Ile Val Arg Gly Thr Ser Val Leu
85 90 95
Gln Gly Asn Glu Lys Leu Thr Phe Gly Thr Gly Thr Arg Leu Thr Ile
100 105 110
Ile Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp
115 120 125
Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser
130 135 140
Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp
145 150 155 160
Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala
165 170 175
Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn
180 185 190
Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser
195 200 205
Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu
210 215 220
Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys
225 230 235 240
Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250 255
<210> 673
<211> 255
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 30 - альфа, цистеин-модифицированный
<400> 673
Asp Ala Lys Thr Thr Gln Pro Pro Ser Met Asp Cys Ala Glu Gly Arg
1 5 10 15
Ala Ala Asn Leu Pro Cys Asn His Ser Thr Ile Ser Gly Asn Glu Tyr
20 25 30
Val Tyr Trp Tyr Arg Gln Ile His Ser Gln Gly Pro Gln Tyr Ile Ile
35 40 45
His Gly Leu Lys Asn Asn Glu Thr Asn Glu Met Ala Ser Leu Ile Ile
50 55 60
Thr Glu Asp Arg Lys Ser Ser Thr Leu Ile Leu Pro His Ala Thr Leu
65 70 75 80
Arg Asp Thr Ala Val Tyr Tyr Cys Ile Val Arg Gly Thr Ser Val Leu
85 90 95
Gln Gly Asn Glu Lys Leu Thr Phe Gly Thr Gly Thr Arg Leu Thr Ile
100 105 110
Ile Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp
115 120 125
Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser
130 135 140
Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp
145 150 155 160
Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala
165 170 175
Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn
180 185 190
Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser
195 200 205
Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu
210 215 220
Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys
225 230 235 240
Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250 255
<210> 674
<211> 272
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 30 - альфа нативный
<400> 674
Met Arg Leu Val Ala Arg Val Thr Val Phe Leu Thr Phe Gly Thr Ile
1 5 10 15
Ile Asp Ala Lys Thr Thr Gln Pro Pro Ser Met Asp Cys Ala Glu Gly
20 25 30
Arg Ala Ala Asn Leu Pro Cys Asn His Ser Thr Ile Ser Gly Asn Glu
35 40 45
Tyr Val Tyr Trp Tyr Arg Gln Ile His Ser Gln Gly Pro Gln Tyr Ile
50 55 60
Ile His Gly Leu Lys Asn Asn Glu Thr Asn Glu Met Ala Ser Leu Ile
65 70 75 80
Ile Thr Glu Asp Arg Lys Ser Ser Thr Leu Ile Leu Pro His Ala Thr
85 90 95
Leu Arg Asp Thr Ala Val Tyr Tyr Cys Ile Val Arg Gly Thr Ser Val
100 105 110
Leu Gln Gly Asn Glu Lys Leu Thr Phe Gly Thr Gly Thr Arg Leu Thr
115 120 125
Ile Ile Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg
130 135 140
Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp
145 150 155 160
Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr
165 170 175
Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser
180 185 190
Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe
195 200 205
Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser
210 215 220
Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn
225 230 235 240
Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu
245 250 255
Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
260 265 270
<210> 675
<211> 272
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 30 - альфа, цистеин-модифицированный
<400> 675
Met Arg Leu Val Ala Arg Val Thr Val Phe Leu Thr Phe Gly Thr Ile
1 5 10 15
Ile Asp Ala Lys Thr Thr Gln Pro Pro Ser Met Asp Cys Ala Glu Gly
20 25 30
Arg Ala Ala Asn Leu Pro Cys Asn His Ser Thr Ile Ser Gly Asn Glu
35 40 45
Tyr Val Tyr Trp Tyr Arg Gln Ile His Ser Gln Gly Pro Gln Tyr Ile
50 55 60
Ile His Gly Leu Lys Asn Asn Glu Thr Asn Glu Met Ala Ser Leu Ile
65 70 75 80
Ile Thr Glu Asp Arg Lys Ser Ser Thr Leu Ile Leu Pro His Ala Thr
85 90 95
Leu Arg Asp Thr Ala Val Tyr Tyr Cys Ile Val Arg Gly Thr Ser Val
100 105 110
Leu Gln Gly Asn Glu Lys Leu Thr Phe Gly Thr Gly Thr Arg Leu Thr
115 120 125
Ile Ile Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg
130 135 140
Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp
145 150 155 160
Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr
165 170 175
Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser
180 185 190
Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe
195 200 205
Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser
210 215 220
Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn
225 230 235 240
Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu
245 250 255
Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
260 265 270
<210> 676
<211> 114
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 30 альфа, вариабельная область
<400> 676
Asp Ala Lys Thr Thr Gln Pro Pro Ser Met Asp Cys Ala Glu Gly Arg
1 5 10 15
Ala Ala Asn Leu Pro Cys Asn His Ser Thr Ile Ser Gly Asn Glu Tyr
20 25 30
Val Tyr Trp Tyr Arg Gln Ile His Ser Gln Gly Pro Gln Tyr Ile Ile
35 40 45
His Gly Leu Lys Asn Asn Glu Thr Asn Glu Met Ala Ser Leu Ile Ile
50 55 60
Thr Glu Asp Arg Lys Ser Ser Thr Leu Ile Leu Pro His Ala Thr Leu
65 70 75 80
Arg Asp Thr Ala Val Tyr Tyr Cys Ile Val Arg Gly Thr Ser Val Leu
85 90 95
Gln Gly Asn Glu Lys Leu Thr Phe Gly Thr Gly Thr Arg Leu Thr Ile
100 105 110
Ile Pro
<210> 677
<211> 7
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 30 альфа CDR1
<400> 677
Thr Ile Ser Gly Asn Glu Tyr
1 5
<210> 678
<211> 5
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 30 альфа CDR2
<400> 678
Gly Leu Lys Asn Asn
1 5
<210> 679
<211> 15
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 30 альфа CDR3
<400> 679
Ile Val Arg Gly Thr Ser Val Leu Gln Gly Asn Glu Lys Leu Thr
1 5 10 15
<210> 680
<211> 17
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 30 альфа, Сигнальный пептид
<400> 680
Met Arg Leu Val Ala Arg Val Thr Val Phe Leu Thr Phe Gly Thr Ile
1 5 10 15
Ile
<210> 681
<211> 292
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 30 - бета нативный
<400> 681
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Ser Ser Arg
85 90 95
Gln Pro Ser Ser Gly Asn Thr Ile Tyr Phe Gly Glu Gly Ser Trp Leu
100 105 110
Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val
115 120 125
Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu
130 135 140
Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp
145 150 155 160
Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln
165 170 175
Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser
180 185 190
Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His
195 200 205
Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp
210 215 220
Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala
225 230 235 240
Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly
245 250 255
Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr
260 265 270
Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys
275 280 285
Arg Lys Asp Phe
290
<210> 682
<211> 292
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 30 - бета, цистеин-модифицированный
<400> 682
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Ser Ser Arg
85 90 95
Gln Pro Ser Ser Gly Asn Thr Ile Tyr Phe Gly Glu Gly Ser Trp Leu
100 105 110
Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val
115 120 125
Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu
130 135 140
Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp
145 150 155 160
Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln
165 170 175
Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser
180 185 190
Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His
195 200 205
Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp
210 215 220
Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala
225 230 235 240
Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly
245 250 255
Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr
260 265 270
Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys
275 280 285
Arg Lys Asp Phe
290
<210> 683
<211> 311
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 30 - бета нативный
<400> 683
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ser Ser Arg Gln Pro Ser Ser Gly Asn Thr Ile Tyr Phe Gly Glu Gly
115 120 125
Ser Trp Leu Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu
130 135 140
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
145 150 155 160
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu
165 170 175
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr
180 185 190
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
195 200 205
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
210 215 220
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
225 230 235 240
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
245 250 255
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr
260 265 270
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
275 280 285
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
290 295 300
Met Val Lys Arg Lys Asp Phe
305 310
<210> 684
<211> 311
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 30 - бета, цистеин-модифицированный
<400> 684
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ser Ser Arg Gln Pro Ser Ser Gly Asn Thr Ile Tyr Phe Gly Glu Gly
115 120 125
Ser Trp Leu Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu
130 135 140
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
145 150 155 160
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu
165 170 175
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr
180 185 190
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
195 200 205
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
210 215 220
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
225 230 235 240
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
245 250 255
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr
260 265 270
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
275 280 285
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
290 295 300
Met Val Lys Arg Lys Asp Phe
305 310
<210> 685
<211> 115
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 30 бета, вариабельная область
<400> 685
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Ser Ser Arg
85 90 95
Gln Pro Ser Ser Gly Asn Thr Ile Tyr Phe Gly Glu Gly Ser Trp Leu
100 105 110
Thr Val Val
115
<210> 686
<211> 13
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 30 бета CDR3
<400> 686
Ala Ser Ser Arg Gln Pro Ser Ser Gly Asn Thr Ile Tyr
1 5 10
<210> 687
<211> 253
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 31 - альфа нативный
<400> 687
Asp Gln Gln Val Lys Gln Asn Ser Pro Ser Leu Ser Val Gln Glu Gly
1 5 10 15
Arg Ile Ser Ile Leu Asn Cys Asp Tyr Thr Asn Ser Met Phe Asp Tyr
20 25 30
Phe Leu Trp Tyr Lys Lys Tyr Pro Ala Glu Gly Pro Thr Phe Leu Ile
35 40 45
Ser Ile Ser Ser Ile Lys Asp Lys Asn Glu Asp Gly Arg Phe Thr Val
50 55 60
Phe Leu Asn Lys Ser Ala Lys His Leu Ser Leu His Ile Val Pro Ser
65 70 75 80
Gln Pro Gly Asp Ser Ala Val Tyr Phe Cys Ala Ala Ser Glu Arg Gly
85 90 95
Thr Tyr Lys Tyr Ile Phe Gly Thr Gly Thr Arg Leu Lys Val Leu Ala
100 105 110
Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys
115 120 125
Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr
130 135 140
Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr
145 150 155 160
Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala
165 170 175
Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser
180 185 190
Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp
195 200 205
Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe
210 215 220
Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala
225 230 235 240
Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 688
<211> 253
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 31 - альфа, цистеин-модифицированный
<400> 688
Asp Gln Gln Val Lys Gln Asn Ser Pro Ser Leu Ser Val Gln Glu Gly
1 5 10 15
Arg Ile Ser Ile Leu Asn Cys Asp Tyr Thr Asn Ser Met Phe Asp Tyr
20 25 30
Phe Leu Trp Tyr Lys Lys Tyr Pro Ala Glu Gly Pro Thr Phe Leu Ile
35 40 45
Ser Ile Ser Ser Ile Lys Asp Lys Asn Glu Asp Gly Arg Phe Thr Val
50 55 60
Phe Leu Asn Lys Ser Ala Lys His Leu Ser Leu His Ile Val Pro Ser
65 70 75 80
Gln Pro Gly Asp Ser Ala Val Tyr Phe Cys Ala Ala Ser Glu Arg Gly
85 90 95
Thr Tyr Lys Tyr Ile Phe Gly Thr Gly Thr Arg Leu Lys Val Leu Ala
100 105 110
Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys
115 120 125
Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr
130 135 140
Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys
145 150 155 160
Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala
165 170 175
Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser
180 185 190
Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp
195 200 205
Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe
210 215 220
Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala
225 230 235 240
Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 689
<211> 279
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 31 - альфа нативный
<400> 689
Met Ala Met Leu Leu Gly Ala Ser Val Leu Ile Leu Trp Leu Gln Pro
1 5 10 15
Asp Trp Val Asn Ser Gln Gln Lys Asn Asp Asp Gln Gln Val Lys Gln
20 25 30
Asn Ser Pro Ser Leu Ser Val Gln Glu Gly Arg Ile Ser Ile Leu Asn
35 40 45
Cys Asp Tyr Thr Asn Ser Met Phe Asp Tyr Phe Leu Trp Tyr Lys Lys
50 55 60
Tyr Pro Ala Glu Gly Pro Thr Phe Leu Ile Ser Ile Ser Ser Ile Lys
65 70 75 80
Asp Lys Asn Glu Asp Gly Arg Phe Thr Val Phe Leu Asn Lys Ser Ala
85 90 95
Lys His Leu Ser Leu His Ile Val Pro Ser Gln Pro Gly Asp Ser Ala
100 105 110
Val Tyr Phe Cys Ala Ala Ser Glu Arg Gly Thr Tyr Lys Tyr Ile Phe
115 120 125
Gly Thr Gly Thr Arg Leu Lys Val Leu Ala Asn Ile Gln Asn Pro Asp
130 135 140
Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val
145 150 155 160
Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys
165 170 175
Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser
180 185 190
Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp
195 200 205
Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr
210 215 220
Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys
225 230 235 240
Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile
245 250 255
Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met
260 265 270
Thr Leu Arg Leu Trp Ser Ser
275
<210> 690
<211> 279
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 31 - альфа, цистеин-модифицированный
<400> 690
Met Ala Met Leu Leu Gly Ala Ser Val Leu Ile Leu Trp Leu Gln Pro
1 5 10 15
Asp Trp Val Asn Ser Gln Gln Lys Asn Asp Asp Gln Gln Val Lys Gln
20 25 30
Asn Ser Pro Ser Leu Ser Val Gln Glu Gly Arg Ile Ser Ile Leu Asn
35 40 45
Cys Asp Tyr Thr Asn Ser Met Phe Asp Tyr Phe Leu Trp Tyr Lys Lys
50 55 60
Tyr Pro Ala Glu Gly Pro Thr Phe Leu Ile Ser Ile Ser Ser Ile Lys
65 70 75 80
Asp Lys Asn Glu Asp Gly Arg Phe Thr Val Phe Leu Asn Lys Ser Ala
85 90 95
Lys His Leu Ser Leu His Ile Val Pro Ser Gln Pro Gly Asp Ser Ala
100 105 110
Val Tyr Phe Cys Ala Ala Ser Glu Arg Gly Thr Tyr Lys Tyr Ile Phe
115 120 125
Gly Thr Gly Thr Arg Leu Lys Val Leu Ala Asn Ile Gln Asn Pro Asp
130 135 140
Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val
145 150 155 160
Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys
165 170 175
Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser
180 185 190
Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp
195 200 205
Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr
210 215 220
Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys
225 230 235 240
Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile
245 250 255
Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met
260 265 270
Thr Leu Arg Leu Trp Ser Ser
275
<210> 691
<211> 112
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 31 альфа, вариабельная область
<400> 691
Asp Gln Gln Val Lys Gln Asn Ser Pro Ser Leu Ser Val Gln Glu Gly
1 5 10 15
Arg Ile Ser Ile Leu Asn Cys Asp Tyr Thr Asn Ser Met Phe Asp Tyr
20 25 30
Phe Leu Trp Tyr Lys Lys Tyr Pro Ala Glu Gly Pro Thr Phe Leu Ile
35 40 45
Ser Ile Ser Ser Ile Lys Asp Lys Asn Glu Asp Gly Arg Phe Thr Val
50 55 60
Phe Leu Asn Lys Ser Ala Lys His Leu Ser Leu His Ile Val Pro Ser
65 70 75 80
Gln Pro Gly Asp Ser Ala Val Tyr Phe Cys Ala Ala Ser Glu Arg Gly
85 90 95
Thr Tyr Lys Tyr Ile Phe Gly Thr Gly Thr Arg Leu Lys Val Leu Ala
100 105 110
<210> 692
<211> 6
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 31 альфа CDR1
<400> 692
Asn Ser Met Phe Asp Tyr
1 5
<210> 693
<211> 7
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 31 альфа CDR2
<400> 693
Ile Ser Ser Ile Lys Asp Lys
1 5
<210> 694
<211> 11
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 31 альфа CDR3
<400> 694
Ala Ala Ser Glu Arg Gly Thr Tyr Lys Tyr Ile
1 5 10
<210> 695
<211> 26
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 31 альфа, Сигнальный пептид
<400> 695
Met Ala Met Leu Leu Gly Ala Ser Val Leu Ile Leu Trp Leu Gln Pro
1 5 10 15
Asp Trp Val Asn Ser Gln Gln Lys Asn Asp
20 25
<210> 696
<211> 294
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 31 - бета нативный
<400> 696
Gly Ala Gly Val Ser Gln Ser Pro Ser Asn Lys Val Thr Glu Lys Gly
1 5 10 15
Lys Asp Val Glu Leu Arg Cys Asp Pro Ile Ser Gly His Thr Ala Leu
20 25 30
Tyr Trp Tyr Arg Gln Ser Leu Gly Gln Gly Leu Glu Phe Leu Ile Tyr
35 40 45
Phe Gln Gly Asn Ser Ala Pro Asp Lys Ser Gly Leu Pro Ser Asp Arg
50 55 60
Phe Ser Ala Glu Arg Thr Gly Gly Ser Val Ser Thr Leu Thr Ile Gln
65 70 75 80
Arg Thr Gln Gln Glu Asp Ser Ala Val Tyr Leu Cys Ala Ser Ser Arg
85 90 95
Phe Leu Gly Ser Thr Asp Thr Gln Tyr Phe Gly Pro Gly Thr Arg Leu
100 105 110
Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val
115 120 125
Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu
130 135 140
Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp
145 150 155 160
Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln
165 170 175
Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser
180 185 190
Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His
195 200 205
Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp
210 215 220
Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala
225 230 235 240
Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly
245 250 255
Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr
260 265 270
Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys
275 280 285
Arg Lys Asp Ser Arg Gly
290
<210> 697
<211> 294
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 31 - бета, цистеин-модифицированный
<400> 697
Gly Ala Gly Val Ser Gln Ser Pro Ser Asn Lys Val Thr Glu Lys Gly
1 5 10 15
Lys Asp Val Glu Leu Arg Cys Asp Pro Ile Ser Gly His Thr Ala Leu
20 25 30
Tyr Trp Tyr Arg Gln Ser Leu Gly Gln Gly Leu Glu Phe Leu Ile Tyr
35 40 45
Phe Gln Gly Asn Ser Ala Pro Asp Lys Ser Gly Leu Pro Ser Asp Arg
50 55 60
Phe Ser Ala Glu Arg Thr Gly Gly Ser Val Ser Thr Leu Thr Ile Gln
65 70 75 80
Arg Thr Gln Gln Glu Asp Ser Ala Val Tyr Leu Cys Ala Ser Ser Arg
85 90 95
Phe Leu Gly Ser Thr Asp Thr Gln Tyr Phe Gly Pro Gly Thr Arg Leu
100 105 110
Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val
115 120 125
Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu
130 135 140
Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp
145 150 155 160
Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln
165 170 175
Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser
180 185 190
Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His
195 200 205
Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp
210 215 220
Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala
225 230 235 240
Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly
245 250 255
Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr
260 265 270
Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys
275 280 285
Arg Lys Asp Ser Arg Gly
290
<210> 698
<211> 313
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 31 - бета нативный
<400> 698
Met Gly Thr Arg Leu Leu Phe Trp Val Ala Phe Cys Leu Leu Gly Ala
1 5 10 15
Asp His Thr Gly Ala Gly Val Ser Gln Ser Pro Ser Asn Lys Val Thr
20 25 30
Glu Lys Gly Lys Asp Val Glu Leu Arg Cys Asp Pro Ile Ser Gly His
35 40 45
Thr Ala Leu Tyr Trp Tyr Arg Gln Ser Leu Gly Gln Gly Leu Glu Phe
50 55 60
Leu Ile Tyr Phe Gln Gly Asn Ser Ala Pro Asp Lys Ser Gly Leu Pro
65 70 75 80
Ser Asp Arg Phe Ser Ala Glu Arg Thr Gly Gly Ser Val Ser Thr Leu
85 90 95
Thr Ile Gln Arg Thr Gln Gln Glu Asp Ser Ala Val Tyr Leu Cys Ala
100 105 110
Ser Ser Arg Phe Leu Gly Ser Thr Asp Thr Gln Tyr Phe Gly Pro Gly
115 120 125
Thr Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu
130 135 140
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
145 150 155 160
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu
165 170 175
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr
180 185 190
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
195 200 205
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
210 215 220
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
225 230 235 240
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
245 250 255
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr
260 265 270
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
275 280 285
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
290 295 300
Met Val Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 699
<211> 313
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 31 - бета, цистеин-модифицированный
<400> 699
Met Gly Thr Arg Leu Leu Phe Trp Val Ala Phe Cys Leu Leu Gly Ala
1 5 10 15
Asp His Thr Gly Ala Gly Val Ser Gln Ser Pro Ser Asn Lys Val Thr
20 25 30
Glu Lys Gly Lys Asp Val Glu Leu Arg Cys Asp Pro Ile Ser Gly His
35 40 45
Thr Ala Leu Tyr Trp Tyr Arg Gln Ser Leu Gly Gln Gly Leu Glu Phe
50 55 60
Leu Ile Tyr Phe Gln Gly Asn Ser Ala Pro Asp Lys Ser Gly Leu Pro
65 70 75 80
Ser Asp Arg Phe Ser Ala Glu Arg Thr Gly Gly Ser Val Ser Thr Leu
85 90 95
Thr Ile Gln Arg Thr Gln Gln Glu Asp Ser Ala Val Tyr Leu Cys Ala
100 105 110
Ser Ser Arg Phe Leu Gly Ser Thr Asp Thr Gln Tyr Phe Gly Pro Gly
115 120 125
Thr Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu
130 135 140
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
145 150 155 160
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu
165 170 175
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr
180 185 190
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
195 200 205
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
210 215 220
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
225 230 235 240
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
245 250 255
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr
260 265 270
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
275 280 285
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
290 295 300
Met Val Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 700
<211> 115
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 31 бета, вариабельная область
<400> 700
Gly Ala Gly Val Ser Gln Ser Pro Ser Asn Lys Val Thr Glu Lys Gly
1 5 10 15
Lys Asp Val Glu Leu Arg Cys Asp Pro Ile Ser Gly His Thr Ala Leu
20 25 30
Tyr Trp Tyr Arg Gln Ser Leu Gly Gln Gly Leu Glu Phe Leu Ile Tyr
35 40 45
Phe Gln Gly Asn Ser Ala Pro Asp Lys Ser Gly Leu Pro Ser Asp Arg
50 55 60
Phe Ser Ala Glu Arg Thr Gly Gly Ser Val Ser Thr Leu Thr Ile Gln
65 70 75 80
Arg Thr Gln Gln Glu Asp Ser Ala Val Tyr Leu Cys Ala Ser Ser Arg
85 90 95
Phe Leu Gly Ser Thr Asp Thr Gln Tyr Phe Gly Pro Gly Thr Arg Leu
100 105 110
Thr Val Leu
115
<210> 701
<211> 5
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 31/ TCR 45/ TCR 46 бета CDR1
<400> 701
Ser Gly His Thr Ala
1 5
<210> 702
<211> 6
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 31/ TCR 45 бета CDR2
<400> 702
Phe Gln Gly Asn Ser Ala
1 5
<210> 703
<211> 13
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 31 бета CDR3
<400> 703
Ala Ser Ser Arg Phe Leu Gly Ser Thr Asp Thr Gln Tyr
1 5 10
<210> 704
<211> 19
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 31/ TCR 32 бета, Сигнальный пептид
<400> 704
Met Gly Thr Arg Leu Leu Phe Trp Val Ala Phe Cys Leu Leu Gly Ala
1 5 10 15
Asp His Thr
<210> 705
<211> 250
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 32 - альфа нативный
<400> 705
Thr Gln Leu Leu Glu Gln Ser Pro Gln Phe Leu Ser Ile Gln Glu Gly
1 5 10 15
Glu Asn Leu Thr Val Tyr Cys Asn Ser Ser Ser Val Phe Ser Ser Leu
20 25 30
Gln Trp Tyr Arg Gln Glu Pro Gly Glu Gly Pro Val Leu Leu Val Thr
35 40 45
Val Val Thr Gly Gly Glu Val Lys Lys Leu Lys Arg Leu Thr Phe Gln
50 55 60
Phe Gly Asp Ala Arg Lys Asp Ser Ser Leu His Ile Thr Ala Ala Gln
65 70 75 80
Pro Gly Asp Thr Gly Leu Tyr Leu Cys Ala Gly Gly Ser Asn Tyr Lys
85 90 95
Leu Thr Phe Gly Lys Gly Thr Leu Leu Thr Val Asn Pro Asn Ile Gln
100 105 110
Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp
115 120 125
Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser
130 135 140
Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr Val Leu Asp
145 150 155 160
Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn
165 170 175
Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro
180 185 190
Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu
195 200 205
Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu
210 215 220
Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn
225 230 235 240
Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 706
<211> 250
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 32 - альфа, цистеин-модифицированный
<400> 706
Thr Gln Leu Leu Glu Gln Ser Pro Gln Phe Leu Ser Ile Gln Glu Gly
1 5 10 15
Glu Asn Leu Thr Val Tyr Cys Asn Ser Ser Ser Val Phe Ser Ser Leu
20 25 30
Gln Trp Tyr Arg Gln Glu Pro Gly Glu Gly Pro Val Leu Leu Val Thr
35 40 45
Val Val Thr Gly Gly Glu Val Lys Lys Leu Lys Arg Leu Thr Phe Gln
50 55 60
Phe Gly Asp Ala Arg Lys Asp Ser Ser Leu His Ile Thr Ala Ala Gln
65 70 75 80
Pro Gly Asp Thr Gly Leu Tyr Leu Cys Ala Gly Gly Ser Asn Tyr Lys
85 90 95
Leu Thr Phe Gly Lys Gly Thr Leu Leu Thr Val Asn Pro Asn Ile Gln
100 105 110
Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp
115 120 125
Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser
130 135 140
Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys Val Leu Asp
145 150 155 160
Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn
165 170 175
Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro
180 185 190
Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu
195 200 205
Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu
210 215 220
Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn
225 230 235 240
Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 707
<211> 268
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 32 - альфа нативный
<400> 707
Met Val Leu Lys Phe Ser Val Ser Ile Leu Trp Ile Gln Leu Ala Trp
1 5 10 15
Val Ser Thr Gln Leu Leu Glu Gln Ser Pro Gln Phe Leu Ser Ile Gln
20 25 30
Glu Gly Glu Asn Leu Thr Val Tyr Cys Asn Ser Ser Ser Val Phe Ser
35 40 45
Ser Leu Gln Trp Tyr Arg Gln Glu Pro Gly Glu Gly Pro Val Leu Leu
50 55 60
Val Thr Val Val Thr Gly Gly Glu Val Lys Lys Leu Lys Arg Leu Thr
65 70 75 80
Phe Gln Phe Gly Asp Ala Arg Lys Asp Ser Ser Leu His Ile Thr Ala
85 90 95
Ala Gln Pro Gly Asp Thr Gly Leu Tyr Leu Cys Ala Gly Gly Ser Asn
100 105 110
Tyr Lys Leu Thr Phe Gly Lys Gly Thr Leu Leu Thr Val Asn Pro Asn
115 120 125
Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser
130 135 140
Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn
145 150 155 160
Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr Val
165 170 175
Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp
180 185 190
Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile
195 200 205
Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val
210 215 220
Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln
225 230 235 240
Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly
245 250 255
Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
260 265
<210> 708
<211> 268
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 32 - альфа, цистеин-модифицированный
<400> 708
Met Val Leu Lys Phe Ser Val Ser Ile Leu Trp Ile Gln Leu Ala Trp
1 5 10 15
Val Ser Thr Gln Leu Leu Glu Gln Ser Pro Gln Phe Leu Ser Ile Gln
20 25 30
Glu Gly Glu Asn Leu Thr Val Tyr Cys Asn Ser Ser Ser Val Phe Ser
35 40 45
Ser Leu Gln Trp Tyr Arg Gln Glu Pro Gly Glu Gly Pro Val Leu Leu
50 55 60
Val Thr Val Val Thr Gly Gly Glu Val Lys Lys Leu Lys Arg Leu Thr
65 70 75 80
Phe Gln Phe Gly Asp Ala Arg Lys Asp Ser Ser Leu His Ile Thr Ala
85 90 95
Ala Gln Pro Gly Asp Thr Gly Leu Tyr Leu Cys Ala Gly Gly Ser Asn
100 105 110
Tyr Lys Leu Thr Phe Gly Lys Gly Thr Leu Leu Thr Val Asn Pro Asn
115 120 125
Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser
130 135 140
Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn
145 150 155 160
Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys Val
165 170 175
Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp
180 185 190
Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile
195 200 205
Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val
210 215 220
Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln
225 230 235 240
Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly
245 250 255
Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
260 265
<210> 709
<211> 109
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 32 альфа, вариабельная область
<400> 709
Thr Gln Leu Leu Glu Gln Ser Pro Gln Phe Leu Ser Ile Gln Glu Gly
1 5 10 15
Glu Asn Leu Thr Val Tyr Cys Asn Ser Ser Ser Val Phe Ser Ser Leu
20 25 30
Gln Trp Tyr Arg Gln Glu Pro Gly Glu Gly Pro Val Leu Leu Val Thr
35 40 45
Val Val Thr Gly Gly Glu Val Lys Lys Leu Lys Arg Leu Thr Phe Gln
50 55 60
Phe Gly Asp Ala Arg Lys Asp Ser Ser Leu His Ile Thr Ala Ala Gln
65 70 75 80
Pro Gly Asp Thr Gly Leu Tyr Leu Cys Ala Gly Gly Ser Asn Tyr Lys
85 90 95
Leu Thr Phe Gly Lys Gly Thr Leu Leu Thr Val Asn Pro
100 105
<210> 710
<211> 5
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 32/ TCR 52 альфа CDR1
<400> 710
Ser Val Phe Ser Ser
1 5
<210> 711
<211> 7
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 32/ TCR 52 альфа CDR2
<400> 711
Val Val Thr Gly Gly Glu Val
1 5
<210> 712
<211> 9
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 32 альфа CDR3
<400> 712
Ala Gly Gly Ser Asn Tyr Lys Leu Thr
1 5
<210> 713
<211> 18
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 32/ TCR 52 альфа, Сигнальный пептид
<400> 713
Met Val Leu Lys Phe Ser Val Ser Ile Leu Trp Ile Gln Leu Ala Trp
1 5 10 15
Val Ser
<210> 714
<211> 293
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 32 - бета нативный
<400> 714
Asp Ser Gly Val Thr Gln Thr Pro Lys His Leu Ile Thr Ala Thr Gly
1 5 10 15
Gln Arg Val Thr Leu Arg Cys Ser Pro Arg Ser Gly Asp Leu Ser Val
20 25 30
Tyr Trp Tyr Gln Gln Ser Leu Asp Gln Gly Leu Gln Phe Leu Ile Gln
35 40 45
Tyr Tyr Asn Gly Glu Glu Arg Ala Lys Gly Asn Ile Leu Glu Arg Phe
50 55 60
Ser Ala Gln Gln Phe Pro Asp Leu His Ser Glu Leu Asn Leu Ser Ser
65 70 75 80
Leu Glu Leu Gly Asp Ser Ala Leu Tyr Phe Cys Ala Ser Ser Val Gly
85 90 95
Gly Asp His Ser Asp Glu Gln Phe Phe Gly Pro Gly Thr Arg Leu Thr
100 105 110
Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe
115 120 125
Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val
130 135 140
Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp
145 150 155 160
Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro
165 170 175
Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser
180 185 190
Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe
195 200 205
Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr
210 215 220
Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp
225 230 235 240
Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val
245 250 255
Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu
260 265 270
Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg
275 280 285
Lys Asp Ser Arg Gly
290
<210> 715
<211> 293
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 32 - бета, цистеин-модифицированный
<400> 715
Asp Ser Gly Val Thr Gln Thr Pro Lys His Leu Ile Thr Ala Thr Gly
1 5 10 15
Gln Arg Val Thr Leu Arg Cys Ser Pro Arg Ser Gly Asp Leu Ser Val
20 25 30
Tyr Trp Tyr Gln Gln Ser Leu Asp Gln Gly Leu Gln Phe Leu Ile Gln
35 40 45
Tyr Tyr Asn Gly Glu Glu Arg Ala Lys Gly Asn Ile Leu Glu Arg Phe
50 55 60
Ser Ala Gln Gln Phe Pro Asp Leu His Ser Glu Leu Asn Leu Ser Ser
65 70 75 80
Leu Glu Leu Gly Asp Ser Ala Leu Tyr Phe Cys Ala Ser Ser Val Gly
85 90 95
Gly Asp His Ser Asp Glu Gln Phe Phe Gly Pro Gly Thr Arg Leu Thr
100 105 110
Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe
115 120 125
Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val
130 135 140
Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp
145 150 155 160
Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro
165 170 175
Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser
180 185 190
Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe
195 200 205
Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr
210 215 220
Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp
225 230 235 240
Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val
245 250 255
Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu
260 265 270
Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg
275 280 285
Lys Asp Ser Arg Gly
290
<210> 716
<211> 312
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 32 - бета нативный
<400> 716
Met Gly Phe Arg Leu Leu Cys Cys Val Ala Phe Cys Leu Leu Gly Ala
1 5 10 15
Gly Pro Val Asp Ser Gly Val Thr Gln Thr Pro Lys His Leu Ile Thr
20 25 30
Ala Thr Gly Gln Arg Val Thr Leu Arg Cys Ser Pro Arg Ser Gly Asp
35 40 45
Leu Ser Val Tyr Trp Tyr Gln Gln Ser Leu Asp Gln Gly Leu Gln Phe
50 55 60
Leu Ile Gln Tyr Tyr Asn Gly Glu Glu Arg Ala Lys Gly Asn Ile Leu
65 70 75 80
Glu Arg Phe Ser Ala Gln Gln Phe Pro Asp Leu His Ser Glu Leu Asn
85 90 95
Leu Ser Ser Leu Glu Leu Gly Asp Ser Ala Leu Tyr Phe Cys Ala Ser
100 105 110
Ser Val Gly Gly Asp His Ser Asp Glu Gln Phe Phe Gly Pro Gly Thr
115 120 125
Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 717
<211> 312
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 32 - бета, цистеин-модифицированный
<400> 717
Met Gly Phe Arg Leu Leu Cys Cys Val Ala Phe Cys Leu Leu Gly Ala
1 5 10 15
Gly Pro Val Asp Ser Gly Val Thr Gln Thr Pro Lys His Leu Ile Thr
20 25 30
Ala Thr Gly Gln Arg Val Thr Leu Arg Cys Ser Pro Arg Ser Gly Asp
35 40 45
Leu Ser Val Tyr Trp Tyr Gln Gln Ser Leu Asp Gln Gly Leu Gln Phe
50 55 60
Leu Ile Gln Tyr Tyr Asn Gly Glu Glu Arg Ala Lys Gly Asn Ile Leu
65 70 75 80
Glu Arg Phe Ser Ala Gln Gln Phe Pro Asp Leu His Ser Glu Leu Asn
85 90 95
Leu Ser Ser Leu Glu Leu Gly Asp Ser Ala Leu Tyr Phe Cys Ala Ser
100 105 110
Ser Val Gly Gly Asp His Ser Asp Glu Gln Phe Phe Gly Pro Gly Thr
115 120 125
Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 718
<211> 114
<212> Белок
<213> Homoe sapiens
<220>
<223> TCR 32 бета, вариабельная область
<400> 718
Asp Ser Gly Val Thr Gln Thr Pro Lys His Leu Ile Thr Ala Thr Gly
1 5 10 15
Gln Arg Val Thr Leu Arg Cys Ser Pro Arg Ser Gly Asp Leu Ser Val
20 25 30
Tyr Trp Tyr Gln Gln Ser Leu Asp Gln Gly Leu Gln Phe Leu Ile Gln
35 40 45
Tyr Tyr Asn Gly Glu Glu Arg Ala Lys Gly Asn Ile Leu Glu Arg Phe
50 55 60
Ser Ala Gln Gln Phe Pro Asp Leu His Ser Glu Leu Asn Leu Ser Ser
65 70 75 80
Leu Glu Leu Gly Asp Ser Ala Leu Tyr Phe Cys Ala Ser Ser Val Gly
85 90 95
Gly Asp His Ser Asp Glu Gln Phe Phe Gly Pro Gly Thr Arg Leu Thr
100 105 110
Val Leu
<210> 719
<211> 5
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 32/ TCR 35/ TCR 44/ TCR 50/ TCR 52 бета CDR1
<400> 719
Ser Gly Asp Leu Ser
1 5
<210> 720
<211> 6
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 32/ TCR 35/ TCR 44/ TCR 50/ TCR 52 бета CDR2
<400> 720
Tyr Tyr Asn Gly Glu Glu
1 5
<210> 721
<211> 13
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 32 бета CDR3
<400> 721
Ala Ser Ser Val Gly Gly Asp His Ser Asp Glu Gln Phe
1 5 10
<210> 722
<211> 254
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 33 - альфа нативный
<400> 722
Gln Lys Glu Val Glu Gln Asn Ser Gly Pro Leu Ser Val Pro Glu Gly
1 5 10 15
Ala Ile Ala Ser Leu Asn Cys Thr Tyr Ser Asp Arg Gly Ser Gln Ser
20 25 30
Phe Phe Trp Tyr Arg Gln Tyr Ser Gly Lys Ser Pro Glu Leu Ile Met
35 40 45
Phe Ile Tyr Ser Asn Gly Asp Lys Glu Asp Gly Arg Phe Thr Ala Gln
50 55 60
Leu Asn Lys Ala Ser Gln Tyr Val Ser Leu Leu Ile Arg Asp Ser Gln
65 70 75 80
Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala Val Asn Ala His His Thr
85 90 95
Gly Gly Phe Lys Thr Ile Phe Gly Ala Gly Thr Arg Leu Phe Val Lys
100 105 110
Ala Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser
115 120 125
Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln
130 135 140
Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys
145 150 155 160
Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val
165 170 175
Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn
180 185 190
Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys
195 200 205
Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn
210 215 220
Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val
225 230 235 240
Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 723
<211> 254
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 33 - альфа, цистеин-модифицированный
<400> 723
Gln Lys Glu Val Glu Gln Asn Ser Gly Pro Leu Ser Val Pro Glu Gly
1 5 10 15
Ala Ile Ala Ser Leu Asn Cys Thr Tyr Ser Asp Arg Gly Ser Gln Ser
20 25 30
Phe Phe Trp Tyr Arg Gln Tyr Ser Gly Lys Ser Pro Glu Leu Ile Met
35 40 45
Phe Ile Tyr Ser Asn Gly Asp Lys Glu Asp Gly Arg Phe Thr Ala Gln
50 55 60
Leu Asn Lys Ala Ser Gln Tyr Val Ser Leu Leu Ile Arg Asp Ser Gln
65 70 75 80
Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala Val Asn Ala His His Thr
85 90 95
Gly Gly Phe Lys Thr Ile Phe Gly Ala Gly Thr Arg Leu Phe Val Lys
100 105 110
Ala Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser
115 120 125
Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln
130 135 140
Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys
145 150 155 160
Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val
165 170 175
Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn
180 185 190
Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys
195 200 205
Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn
210 215 220
Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val
225 230 235 240
Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 724
<211> 276
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 33 - альфа нативный
<400> 724
Met Met Lys Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu
1 5 10 15
Ser Trp Val Trp Ser Gln Gln Lys Glu Val Glu Gln Asn Ser Gly Pro
20 25 30
Leu Ser Val Pro Glu Gly Ala Ile Ala Ser Leu Asn Cys Thr Tyr Ser
35 40 45
Asp Arg Gly Ser Gln Ser Phe Phe Trp Tyr Arg Gln Tyr Ser Gly Lys
50 55 60
Ser Pro Glu Leu Ile Met Phe Ile Tyr Ser Asn Gly Asp Lys Glu Asp
65 70 75 80
Gly Arg Phe Thr Ala Gln Leu Asn Lys Ala Ser Gln Tyr Val Ser Leu
85 90 95
Leu Ile Arg Asp Ser Gln Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala
100 105 110
Val Asn Ala His His Thr Gly Gly Phe Lys Thr Ile Phe Gly Ala Gly
115 120 125
Thr Arg Leu Phe Val Lys Ala Asn Ile Gln Asn Pro Asp Pro Ala Val
130 135 140
Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe
145 150 155 160
Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp
165 170 175
Val Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe
180 185 190
Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys
195 200 205
Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro
210 215 220
Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu
225 230 235 240
Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg
245 250 255
Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg
260 265 270
Leu Trp Ser Ser
275
<210> 725
<211> 276
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 33 - альфа, цистеин-модифицированный
<400> 725
Met Met Lys Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu
1 5 10 15
Ser Trp Val Trp Ser Gln Gln Lys Glu Val Glu Gln Asn Ser Gly Pro
20 25 30
Leu Ser Val Pro Glu Gly Ala Ile Ala Ser Leu Asn Cys Thr Tyr Ser
35 40 45
Asp Arg Gly Ser Gln Ser Phe Phe Trp Tyr Arg Gln Tyr Ser Gly Lys
50 55 60
Ser Pro Glu Leu Ile Met Phe Ile Tyr Ser Asn Gly Asp Lys Glu Asp
65 70 75 80
Gly Arg Phe Thr Ala Gln Leu Asn Lys Ala Ser Gln Tyr Val Ser Leu
85 90 95
Leu Ile Arg Asp Ser Gln Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala
100 105 110
Val Asn Ala His His Thr Gly Gly Phe Lys Thr Ile Phe Gly Ala Gly
115 120 125
Thr Arg Leu Phe Val Lys Ala Asn Ile Gln Asn Pro Asp Pro Ala Val
130 135 140
Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe
145 150 155 160
Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp
165 170 175
Val Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe
180 185 190
Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys
195 200 205
Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro
210 215 220
Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu
225 230 235 240
Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg
245 250 255
Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg
260 265 270
Leu Trp Ser Ser
275
<210> 726
<211> 113
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 33 альфа, вариабельная область
<400> 726
Gln Lys Glu Val Glu Gln Asn Ser Gly Pro Leu Ser Val Pro Glu Gly
1 5 10 15
Ala Ile Ala Ser Leu Asn Cys Thr Tyr Ser Asp Arg Gly Ser Gln Ser
20 25 30
Phe Phe Trp Tyr Arg Gln Tyr Ser Gly Lys Ser Pro Glu Leu Ile Met
35 40 45
Phe Ile Tyr Ser Asn Gly Asp Lys Glu Asp Gly Arg Phe Thr Ala Gln
50 55 60
Leu Asn Lys Ala Ser Gln Tyr Val Ser Leu Leu Ile Arg Asp Ser Gln
65 70 75 80
Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala Val Asn Ala His His Thr
85 90 95
Gly Gly Phe Lys Thr Ile Phe Gly Ala Gly Thr Arg Leu Phe Val Lys
100 105 110
Ala
<210> 727
<211> 6
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 33/ TCR 43/ TCR 47/ TCR 49/ TCR 51 альфа CDR1
<400> 727
Asp Arg Gly Ser Gln Ser
1 5
<210> 728
<211> 6
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 33/ TCR 43/ TCR 47/ TCR 49/ TCR 51 альфа CDR2
<400> 728
Ile Tyr Ser Asn Gly Asp
1 5
<210> 729
<211> 13
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 33 альфа CDR3
<400> 729
Ala Val Asn Ala His His Thr Gly Gly Phe Lys Thr Ile
1 5 10
<210> 730
<211> 22
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 33/ TCR 43/ TCR 47/ TCR 48/ TCR 49/ TCR 51
альфа, Сигнальный пептид
<400> 730
Met Met Lys Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu
1 5 10 15
Ser Trp Val Trp Ser Gln
20
<210> 731
<211> 292
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 33 - бета нативный
<400> 731
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Ser Thr Pro
85 90 95
Arg Asp Thr Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu
115 120 125
Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys
130 135 140
Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val
145 150 155 160
Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro Leu
165 170 175
Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg
180 185 190
Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg
195 200 205
Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln
210 215 220
Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly
225 230 235 240
Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu
245 250 255
Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr
260 265 270
Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys
275 280 285
Asp Ser Arg Gly
290
<210> 732
<211> 292
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 33 - бета, цистеин-модифицированный
<400> 732
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Ser Thr Pro
85 90 95
Arg Asp Thr Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu
115 120 125
Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys
130 135 140
Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val
145 150 155 160
Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro Leu
165 170 175
Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg
180 185 190
Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg
195 200 205
Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln
210 215 220
Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly
225 230 235 240
Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu
245 250 255
Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr
260 265 270
Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys
275 280 285
Asp Ser Arg Gly
290
<210> 733
<211> 311
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 33 - бета нативный
<400> 733
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ser Thr Pro Arg Asp Thr Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 734
<211> 311
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 33 - бета, цистеин-модифицированный
<400> 734
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ser Thr Pro Arg Asp Thr Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 735
<211> 113
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 33 бета, вариабельная область
<400> 735
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Ser Thr Pro
85 90 95
Arg Asp Thr Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Thr
<210> 736
<211> 11
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 33 бета CDR3
<400> 736
Ala Ser Thr Pro Arg Asp Thr Tyr Glu Gln Tyr
1 5 10
<210> 737
<211> 250
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 34 - альфа нативный
<400> 737
Glu Leu Lys Val Glu Gln Asn Pro Leu Phe Leu Ser Met Gln Glu Gly
1 5 10 15
Lys Asn Tyr Thr Ile Tyr Cys Asn Tyr Ser Thr Thr Ser Asp Arg Leu
20 25 30
Tyr Trp Tyr Arg Gln Asp Pro Gly Lys Ser Leu Glu Ser Leu Phe Val
35 40 45
Leu Leu Ser Asn Gly Ala Val Lys Gln Glu Gly Arg Leu Met Ala Ser
50 55 60
Leu Asp Thr Lys Ala Arg Leu Ser Thr Leu His Ile Thr Ala Ala Val
65 70 75 80
His Asp Leu Ser Ala Thr Tyr Phe Cys Ala Val Ser Gly Thr Tyr Lys
85 90 95
Tyr Ile Phe Gly Thr Gly Thr Arg Leu Lys Val Leu Ala Asn Ile Gln
100 105 110
Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp
115 120 125
Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser
130 135 140
Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr Val Leu Asp
145 150 155 160
Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn
165 170 175
Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro
180 185 190
Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu
195 200 205
Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu
210 215 220
Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn
225 230 235 240
Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 738
<211> 250
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 34 - альфа, цистеин-модифицированный
<400> 738
Glu Leu Lys Val Glu Gln Asn Pro Leu Phe Leu Ser Met Gln Glu Gly
1 5 10 15
Lys Asn Tyr Thr Ile Tyr Cys Asn Tyr Ser Thr Thr Ser Asp Arg Leu
20 25 30
Tyr Trp Tyr Arg Gln Asp Pro Gly Lys Ser Leu Glu Ser Leu Phe Val
35 40 45
Leu Leu Ser Asn Gly Ala Val Lys Gln Glu Gly Arg Leu Met Ala Ser
50 55 60
Leu Asp Thr Lys Ala Arg Leu Ser Thr Leu His Ile Thr Ala Ala Val
65 70 75 80
His Asp Leu Ser Ala Thr Tyr Phe Cys Ala Val Ser Gly Thr Tyr Lys
85 90 95
Tyr Ile Phe Gly Thr Gly Thr Arg Leu Lys Val Leu Ala Asn Ile Gln
100 105 110
Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp
115 120 125
Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser
130 135 140
Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys Val Leu Asp
145 150 155 160
Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn
165 170 175
Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro
180 185 190
Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu
195 200 205
Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu
210 215 220
Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn
225 230 235 240
Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 739
<211> 268
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 34 - альфа нативный
<400> 739
Met Lys Lys Leu Leu Ala Met Ile Leu Trp Leu Gln Leu Asp Arg Leu
1 5 10 15
Ser Gly Glu Leu Lys Val Glu Gln Asn Pro Leu Phe Leu Ser Met Gln
20 25 30
Glu Gly Lys Asn Tyr Thr Ile Tyr Cys Asn Tyr Ser Thr Thr Ser Asp
35 40 45
Arg Leu Tyr Trp Tyr Arg Gln Asp Pro Gly Lys Ser Leu Glu Ser Leu
50 55 60
Phe Val Leu Leu Ser Asn Gly Ala Val Lys Gln Glu Gly Arg Leu Met
65 70 75 80
Ala Ser Leu Asp Thr Lys Ala Arg Leu Ser Thr Leu His Ile Thr Ala
85 90 95
Ala Val His Asp Leu Ser Ala Thr Tyr Phe Cys Ala Val Ser Gly Thr
100 105 110
Tyr Lys Tyr Ile Phe Gly Thr Gly Thr Arg Leu Lys Val Leu Ala Asn
115 120 125
Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser
130 135 140
Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn
145 150 155 160
Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr Val
165 170 175
Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp
180 185 190
Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile
195 200 205
Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val
210 215 220
Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln
225 230 235 240
Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly
245 250 255
Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
260 265
<210> 740
<211> 268
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 34 - альфа, цистеин-модифицированный
<400> 740
Met Lys Lys Leu Leu Ala Met Ile Leu Trp Leu Gln Leu Asp Arg Leu
1 5 10 15
Ser Gly Glu Leu Lys Val Glu Gln Asn Pro Leu Phe Leu Ser Met Gln
20 25 30
Glu Gly Lys Asn Tyr Thr Ile Tyr Cys Asn Tyr Ser Thr Thr Ser Asp
35 40 45
Arg Leu Tyr Trp Tyr Arg Gln Asp Pro Gly Lys Ser Leu Glu Ser Leu
50 55 60
Phe Val Leu Leu Ser Asn Gly Ala Val Lys Gln Glu Gly Arg Leu Met
65 70 75 80
Ala Ser Leu Asp Thr Lys Ala Arg Leu Ser Thr Leu His Ile Thr Ala
85 90 95
Ala Val His Asp Leu Ser Ala Thr Tyr Phe Cys Ala Val Ser Gly Thr
100 105 110
Tyr Lys Tyr Ile Phe Gly Thr Gly Thr Arg Leu Lys Val Leu Ala Asn
115 120 125
Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser
130 135 140
Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn
145 150 155 160
Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys Val
165 170 175
Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp
180 185 190
Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile
195 200 205
Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val
210 215 220
Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln
225 230 235 240
Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly
245 250 255
Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
260 265
<210> 741
<211> 109
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 34 альфа, вариабельная область
<400> 741
Glu Leu Lys Val Glu Gln Asn Pro Leu Phe Leu Ser Met Gln Glu Gly
1 5 10 15
Lys Asn Tyr Thr Ile Tyr Cys Asn Tyr Ser Thr Thr Ser Asp Arg Leu
20 25 30
Tyr Trp Tyr Arg Gln Asp Pro Gly Lys Ser Leu Glu Ser Leu Phe Val
35 40 45
Leu Leu Ser Asn Gly Ala Val Lys Gln Glu Gly Arg Leu Met Ala Ser
50 55 60
Leu Asp Thr Lys Ala Arg Leu Ser Thr Leu His Ile Thr Ala Ala Val
65 70 75 80
His Asp Leu Ser Ala Thr Tyr Phe Cys Ala Val Ser Gly Thr Tyr Lys
85 90 95
Tyr Ile Phe Gly Thr Gly Thr Arg Leu Lys Val Leu Ala
100 105
<210> 742
<211> 5
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 34/ TCR 37 альфа CDR1
<400> 742
Thr Thr Ser Asp Arg
1 5
<210> 743
<211> 7
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 34/ TCR 37 альфа CDR2
<400> 743
Leu Leu Ser Asn Gly Ala Val
1 5
<210> 744
<211> 9
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 34 альфа CDR3
<400> 744
Ala Val Ser Gly Thr Tyr Lys Tyr Ile
1 5
<210> 745
<211> 18
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 34/ TCR 37 альфа, Сигнальный пептид
<400> 745
Met Lys Lys Leu Leu Ala Met Ile Leu Trp Leu Gln Leu Asp Arg Leu
1 5 10 15
Ser Gly
<210> 746
<211> 292
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 34 - бета нативный
<400> 746
Glu Thr Gly Val Thr Gln Ser Pro Thr His Leu Ile Lys Thr Arg Gly
1 5 10 15
Gln Gln Val Thr Leu Arg Cys Ser Ser Gln Ser Gly His Asn Thr Val
20 25 30
Ser Trp Tyr Gln Gln Ala Leu Gly Gln Gly Pro Gln Phe Ile Phe Gln
35 40 45
Tyr Tyr Arg Glu Glu Glu Asn Gly Arg Gly Asn Phe Pro Pro Arg Phe
50 55 60
Ser Gly Leu Gln Phe Pro Asn Tyr Ser Ser Glu Leu Asn Val Asn Ala
65 70 75 80
Leu Glu Leu Asp Asp Ser Ala Leu Tyr Leu Cys Ala Ser Ser Ser Tyr
85 90 95
Ala Gly Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu
115 120 125
Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys
130 135 140
Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val
145 150 155 160
Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro Leu
165 170 175
Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg
180 185 190
Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg
195 200 205
Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln
210 215 220
Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly
225 230 235 240
Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu
245 250 255
Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr
260 265 270
Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys
275 280 285
Asp Ser Arg Gly
290
<210> 747
<211> 292
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 34 - бета, цистеин-модифицированный
<400> 747
Glu Thr Gly Val Thr Gln Ser Pro Thr His Leu Ile Lys Thr Arg Gly
1 5 10 15
Gln Gln Val Thr Leu Arg Cys Ser Ser Gln Ser Gly His Asn Thr Val
20 25 30
Ser Trp Tyr Gln Gln Ala Leu Gly Gln Gly Pro Gln Phe Ile Phe Gln
35 40 45
Tyr Tyr Arg Glu Glu Glu Asn Gly Arg Gly Asn Phe Pro Pro Arg Phe
50 55 60
Ser Gly Leu Gln Phe Pro Asn Tyr Ser Ser Glu Leu Asn Val Asn Ala
65 70 75 80
Leu Glu Leu Asp Asp Ser Ala Leu Tyr Leu Cys Ala Ser Ser Ser Tyr
85 90 95
Ala Gly Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu
115 120 125
Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys
130 135 140
Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val
145 150 155 160
Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro Leu
165 170 175
Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg
180 185 190
Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg
195 200 205
Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln
210 215 220
Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly
225 230 235 240
Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu
245 250 255
Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr
260 265 270
Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys
275 280 285
Asp Ser Arg Gly
290
<210> 748
<211> 311
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 34 - бета нативный
<400> 748
Met Gly Pro Gly Leu Leu Cys Trp Ala Leu Leu Cys Leu Leu Gly Ala
1 5 10 15
Gly Ser Val Glu Thr Gly Val Thr Gln Ser Pro Thr His Leu Ile Lys
20 25 30
Thr Arg Gly Gln Gln Val Thr Leu Arg Cys Ser Ser Gln Ser Gly His
35 40 45
Asn Thr Val Ser Trp Tyr Gln Gln Ala Leu Gly Gln Gly Pro Gln Phe
50 55 60
Ile Phe Gln Tyr Tyr Arg Glu Glu Glu Asn Gly Arg Gly Asn Phe Pro
65 70 75 80
Pro Arg Phe Ser Gly Leu Gln Phe Pro Asn Tyr Ser Ser Glu Leu Asn
85 90 95
Val Asn Ala Leu Glu Leu Asp Asp Ser Ala Leu Tyr Leu Cys Ala Ser
100 105 110
Ser Ser Tyr Ala Gly Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 749
<211> 311
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 34 - бета, цистеин-модифицированный
<400> 749
Met Gly Pro Gly Leu Leu Cys Trp Ala Leu Leu Cys Leu Leu Gly Ala
1 5 10 15
Gly Ser Val Glu Thr Gly Val Thr Gln Ser Pro Thr His Leu Ile Lys
20 25 30
Thr Arg Gly Gln Gln Val Thr Leu Arg Cys Ser Ser Gln Ser Gly His
35 40 45
Asn Thr Val Ser Trp Tyr Gln Gln Ala Leu Gly Gln Gly Pro Gln Phe
50 55 60
Ile Phe Gln Tyr Tyr Arg Glu Glu Glu Asn Gly Arg Gly Asn Phe Pro
65 70 75 80
Pro Arg Phe Ser Gly Leu Gln Phe Pro Asn Tyr Ser Ser Glu Leu Asn
85 90 95
Val Asn Ala Leu Glu Leu Asp Asp Ser Ala Leu Tyr Leu Cys Ala Ser
100 105 110
Ser Ser Tyr Ala Gly Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 750
<211> 113
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 34 бета, вариабельная область
<400> 750
Glu Thr Gly Val Thr Gln Ser Pro Thr His Leu Ile Lys Thr Arg Gly
1 5 10 15
Gln Gln Val Thr Leu Arg Cys Ser Ser Gln Ser Gly His Asn Thr Val
20 25 30
Ser Trp Tyr Gln Gln Ala Leu Gly Gln Gly Pro Gln Phe Ile Phe Gln
35 40 45
Tyr Tyr Arg Glu Glu Glu Asn Gly Arg Gly Asn Phe Pro Pro Arg Phe
50 55 60
Ser Gly Leu Gln Phe Pro Asn Tyr Ser Ser Glu Leu Asn Val Asn Ala
65 70 75 80
Leu Glu Leu Asp Asp Ser Ala Leu Tyr Leu Cys Ala Ser Ser Ser Tyr
85 90 95
Ala Gly Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Thr
<210> 751
<211> 5
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 34/ TCR 38 бета CDR1
<400> 751
Ser Gly His Asn Thr
1 5
<210> 752
<211> 6
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 34/ TCR 38 бета CDR2
<400> 752
Tyr Tyr Arg Glu Glu Glu
1 5
<210> 753
<211> 13
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 34 бета CDR3
<400> 753
Ala Ser Ser Ser Tyr Ala Gly Ser Tyr Glu Gln Tyr Phe
1 5 10
<210> 754
<211> 19
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 34 бета, Сигнальный пептид
<400> 754
Met Gly Pro Gly Leu Leu Cys Trp Ala Leu Leu Cys Leu Leu Gly Ala
1 5 10 15
Gly Ser Val
<210> 755
<211> 245
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 35 - альфа нативный
<400> 755
Lys Asp Gln Val Phe Gln Pro Ser Thr Val Ala Ser Ser Glu Gly Ala
1 5 10 15
Val Val Glu Ile Phe Cys Asn His Ser Val Ser Asn Ala Tyr Asn Phe
20 25 30
Phe Trp Tyr Leu His Phe Pro Gly Cys Ala Pro Arg Leu Leu Val Lys
35 40 45
Gly Ser Lys Pro Ser Gln Gln Gly Arg Tyr Asn Met Thr Tyr Glu Arg
50 55 60
Phe Ser Ser Ser Leu Leu Ile Leu Gln Val Arg Glu Ala Asp Ala Ala
65 70 75 80
Val Tyr Tyr Cys Ala Val Ala Gly Asp Tyr Lys Leu Ser Phe Gly Ala
85 90 95
Gly Thr Thr Val Thr Val Arg Ala Asn Ile Gln Asn Pro Asp Pro Ala
100 105 110
Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu
115 120 125
Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser
130 135 140
Asp Val Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp
145 150 155 160
Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala
165 170 175
Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe
180 185 190
Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe
195 200 205
Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe
210 215 220
Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu
225 230 235 240
Arg Leu Trp Ser Ser
245
<210> 756
<211> 245
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 35 - альфа, цистеин-модифицированный
<400> 756
Lys Asp Gln Val Phe Gln Pro Ser Thr Val Ala Ser Ser Glu Gly Ala
1 5 10 15
Val Val Glu Ile Phe Cys Asn His Ser Val Ser Asn Ala Tyr Asn Phe
20 25 30
Phe Trp Tyr Leu His Phe Pro Gly Cys Ala Pro Arg Leu Leu Val Lys
35 40 45
Gly Ser Lys Pro Ser Gln Gln Gly Arg Tyr Asn Met Thr Tyr Glu Arg
50 55 60
Phe Ser Ser Ser Leu Leu Ile Leu Gln Val Arg Glu Ala Asp Ala Ala
65 70 75 80
Val Tyr Tyr Cys Ala Val Ala Gly Asp Tyr Lys Leu Ser Phe Gly Ala
85 90 95
Gly Thr Thr Val Thr Val Arg Ala Asn Ile Gln Asn Pro Asp Pro Ala
100 105 110
Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu
115 120 125
Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser
130 135 140
Asp Val Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp
145 150 155 160
Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala
165 170 175
Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe
180 185 190
Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe
195 200 205
Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe
210 215 220
Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu
225 230 235 240
Arg Leu Trp Ser Ser
245
<210> 757
<211> 270
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 35 - альфа нативный
<400> 757
Met Ala Leu Gln Ser Thr Leu Gly Ala Val Trp Leu Gly Leu Leu Leu
1 5 10 15
Asn Ser Leu Trp Lys Val Ala Glu Ser Lys Asp Gln Val Phe Gln Pro
20 25 30
Ser Thr Val Ala Ser Ser Glu Gly Ala Val Val Glu Ile Phe Cys Asn
35 40 45
His Ser Val Ser Asn Ala Tyr Asn Phe Phe Trp Tyr Leu His Phe Pro
50 55 60
Gly Cys Ala Pro Arg Leu Leu Val Lys Gly Ser Lys Pro Ser Gln Gln
65 70 75 80
Gly Arg Tyr Asn Met Thr Tyr Glu Arg Phe Ser Ser Ser Leu Leu Ile
85 90 95
Leu Gln Val Arg Glu Ala Asp Ala Ala Val Tyr Tyr Cys Ala Val Ala
100 105 110
Gly Asp Tyr Lys Leu Ser Phe Gly Ala Gly Thr Thr Val Thr Val Arg
115 120 125
Ala Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser
130 135 140
Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln
145 150 155 160
Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys
165 170 175
Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val
180 185 190
Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn
195 200 205
Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys
210 215 220
Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn
225 230 235 240
Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val
245 250 255
Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
260 265 270
<210> 758
<211> 270
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 35 - альфа, цистеин-модифицированный
<400> 758
Met Ala Leu Gln Ser Thr Leu Gly Ala Val Trp Leu Gly Leu Leu Leu
1 5 10 15
Asn Ser Leu Trp Lys Val Ala Glu Ser Lys Asp Gln Val Phe Gln Pro
20 25 30
Ser Thr Val Ala Ser Ser Glu Gly Ala Val Val Glu Ile Phe Cys Asn
35 40 45
His Ser Val Ser Asn Ala Tyr Asn Phe Phe Trp Tyr Leu His Phe Pro
50 55 60
Gly Cys Ala Pro Arg Leu Leu Val Lys Gly Ser Lys Pro Ser Gln Gln
65 70 75 80
Gly Arg Tyr Asn Met Thr Tyr Glu Arg Phe Ser Ser Ser Leu Leu Ile
85 90 95
Leu Gln Val Arg Glu Ala Asp Ala Ala Val Tyr Tyr Cys Ala Val Ala
100 105 110
Gly Asp Tyr Lys Leu Ser Phe Gly Ala Gly Thr Thr Val Thr Val Arg
115 120 125
Ala Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser
130 135 140
Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln
145 150 155 160
Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys
165 170 175
Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val
180 185 190
Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn
195 200 205
Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys
210 215 220
Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn
225 230 235 240
Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val
245 250 255
Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
260 265 270
<210> 759
<211> 104
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 35 альфа, вариабельная область
<400> 759
Lys Asp Gln Val Phe Gln Pro Ser Thr Val Ala Ser Ser Glu Gly Ala
1 5 10 15
Val Val Glu Ile Phe Cys Asn His Ser Val Ser Asn Ala Tyr Asn Phe
20 25 30
Phe Trp Tyr Leu His Phe Pro Gly Cys Ala Pro Arg Leu Leu Val Lys
35 40 45
Gly Ser Lys Pro Ser Gln Gln Gly Arg Tyr Asn Met Thr Tyr Glu Arg
50 55 60
Phe Ser Ser Ser Leu Leu Ile Leu Gln Val Arg Glu Ala Asp Ala Ala
65 70 75 80
Val Tyr Tyr Cys Ala Val Ala Gly Asp Tyr Lys Leu Ser Phe Gly Ala
85 90 95
Gly Thr Thr Val Thr Val Arg Ala
100
<210> 760
<211> 6
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 35 альфа CDR1
<400> 760
Val Ser Asn Ala Tyr Asn
1 5
<210> 761
<211> 4
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 35 альфа CDR2
<400> 761
Gly Ser Lys Pro
1
<210> 762
<211> 9
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 35 альфа CDR3
<400> 762
Ala Val Ala Gly Asp Tyr Lys Leu Ser
1 5
<210> 763
<211> 25
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 35 альфа, Сигнальный пептид
<400> 763
Met Ala Leu Gln Ser Thr Leu Gly Ala Val Trp Leu Gly Leu Leu Leu
1 5 10 15
Asn Ser Leu Trp Lys Val Ala Glu Ser
20 25
<210> 764
<211> 294
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 35 - бета нативный
<400> 764
Asp Ser Gly Val Thr Gln Thr Pro Lys His Leu Ile Thr Ala Thr Gly
1 5 10 15
Gln Arg Val Thr Leu Arg Cys Ser Pro Arg Ser Gly Asp Leu Ser Val
20 25 30
Tyr Trp Tyr Gln Gln Ser Leu Asp Gln Gly Leu Gln Phe Leu Ile Gln
35 40 45
Tyr Tyr Asn Gly Glu Glu Arg Ala Lys Gly Asn Ile Leu Glu Arg Phe
50 55 60
Ser Ala Gln Gln Phe Pro Asp Leu His Ser Glu Leu Asn Leu Ser Ser
65 70 75 80
Leu Glu Leu Gly Asp Ser Ala Leu Tyr Phe Cys Ala Ser Thr Thr Ser
85 90 95
Gly Asp Ser Ser Tyr Asn Glu Gln Phe Phe Gly Pro Gly Thr Arg Leu
100 105 110
Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val
115 120 125
Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu
130 135 140
Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp
145 150 155 160
Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln
165 170 175
Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser
180 185 190
Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His
195 200 205
Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp
210 215 220
Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala
225 230 235 240
Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly
245 250 255
Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr
260 265 270
Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys
275 280 285
Arg Lys Asp Ser Arg Gly
290
<210> 765
<211> 294
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 35 - бета, цистеин-модифицированный
<400> 765
Asp Ser Gly Val Thr Gln Thr Pro Lys His Leu Ile Thr Ala Thr Gly
1 5 10 15
Gln Arg Val Thr Leu Arg Cys Ser Pro Arg Ser Gly Asp Leu Ser Val
20 25 30
Tyr Trp Tyr Gln Gln Ser Leu Asp Gln Gly Leu Gln Phe Leu Ile Gln
35 40 45
Tyr Tyr Asn Gly Glu Glu Arg Ala Lys Gly Asn Ile Leu Glu Arg Phe
50 55 60
Ser Ala Gln Gln Phe Pro Asp Leu His Ser Glu Leu Asn Leu Ser Ser
65 70 75 80
Leu Glu Leu Gly Asp Ser Ala Leu Tyr Phe Cys Ala Ser Thr Thr Ser
85 90 95
Gly Asp Ser Ser Tyr Asn Glu Gln Phe Phe Gly Pro Gly Thr Arg Leu
100 105 110
Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val
115 120 125
Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu
130 135 140
Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp
145 150 155 160
Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln
165 170 175
Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser
180 185 190
Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His
195 200 205
Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp
210 215 220
Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala
225 230 235 240
Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly
245 250 255
Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr
260 265 270
Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys
275 280 285
Arg Lys Asp Ser Arg Gly
290
<210> 766
<211> 313
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 35 - бета нативный
<400> 766
Met Gly Phe Arg Leu Leu Cys Cys Val Ala Phe Cys Leu Leu Gly Ala
1 5 10 15
Gly Pro Val Asp Ser Gly Val Thr Gln Thr Pro Lys His Leu Ile Thr
20 25 30
Ala Thr Gly Gln Arg Val Thr Leu Arg Cys Ser Pro Arg Ser Gly Asp
35 40 45
Leu Ser Val Tyr Trp Tyr Gln Gln Ser Leu Asp Gln Gly Leu Gln Phe
50 55 60
Leu Ile Gln Tyr Tyr Asn Gly Glu Glu Arg Ala Lys Gly Asn Ile Leu
65 70 75 80
Glu Arg Phe Ser Ala Gln Gln Phe Pro Asp Leu His Ser Glu Leu Asn
85 90 95
Leu Ser Ser Leu Glu Leu Gly Asp Ser Ala Leu Tyr Phe Cys Ala Ser
100 105 110
Thr Thr Ser Gly Asp Ser Ser Tyr Asn Glu Gln Phe Phe Gly Pro Gly
115 120 125
Thr Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu
130 135 140
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
145 150 155 160
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu
165 170 175
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr
180 185 190
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
195 200 205
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
210 215 220
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
225 230 235 240
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
245 250 255
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr
260 265 270
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
275 280 285
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
290 295 300
Met Val Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 767
<211> 313
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 35 - бета, цистеин-модифицированный
<400> 767
Met Gly Phe Arg Leu Leu Cys Cys Val Ala Phe Cys Leu Leu Gly Ala
1 5 10 15
Gly Pro Val Asp Ser Gly Val Thr Gln Thr Pro Lys His Leu Ile Thr
20 25 30
Ala Thr Gly Gln Arg Val Thr Leu Arg Cys Ser Pro Arg Ser Gly Asp
35 40 45
Leu Ser Val Tyr Trp Tyr Gln Gln Ser Leu Asp Gln Gly Leu Gln Phe
50 55 60
Leu Ile Gln Tyr Tyr Asn Gly Glu Glu Arg Ala Lys Gly Asn Ile Leu
65 70 75 80
Glu Arg Phe Ser Ala Gln Gln Phe Pro Asp Leu His Ser Glu Leu Asn
85 90 95
Leu Ser Ser Leu Glu Leu Gly Asp Ser Ala Leu Tyr Phe Cys Ala Ser
100 105 110
Thr Thr Ser Gly Asp Ser Ser Tyr Asn Glu Gln Phe Phe Gly Pro Gly
115 120 125
Thr Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu
130 135 140
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
145 150 155 160
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu
165 170 175
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr
180 185 190
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
195 200 205
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
210 215 220
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
225 230 235 240
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
245 250 255
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr
260 265 270
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
275 280 285
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
290 295 300
Met Val Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 768
<211> 115
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 35 бета, вариабельная область
<400> 768
Asp Ser Gly Val Thr Gln Thr Pro Lys His Leu Ile Thr Ala Thr Gly
1 5 10 15
Gln Arg Val Thr Leu Arg Cys Ser Pro Arg Ser Gly Asp Leu Ser Val
20 25 30
Tyr Trp Tyr Gln Gln Ser Leu Asp Gln Gly Leu Gln Phe Leu Ile Gln
35 40 45
Tyr Tyr Asn Gly Glu Glu Arg Ala Lys Gly Asn Ile Leu Glu Arg Phe
50 55 60
Ser Ala Gln Gln Phe Pro Asp Leu His Ser Glu Leu Asn Leu Ser Ser
65 70 75 80
Leu Glu Leu Gly Asp Ser Ala Leu Tyr Phe Cys Ala Ser Thr Thr Ser
85 90 95
Gly Asp Ser Ser Tyr Asn Glu Gln Phe Phe Gly Pro Gly Thr Arg Leu
100 105 110
Thr Val Leu
115
<210> 769
<211> 14
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 35 бета CDR3
<400> 769
Ala Ser Thr Thr Ser Gly Asp Ser Ser Tyr Asn Glu Gln Phe
1 5 10
<210> 770
<211> 19
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 35/ TCR 44/ TCR 50/ TCR 52 бета, Сигнальный пептид
<400> 770
Met Gly Phe Arg Leu Leu Cys Cys Val Ala Phe Cys Leu Leu Gly Ala
1 5 10 15
Gly Pro Val
<210> 771
<211> 252
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 36 - альфа нативный
<400> 771
Arg Lys Glu Val Glu Gln Asp Pro Gly Pro Phe Asn Val Pro Glu Gly
1 5 10 15
Ala Thr Val Ala Phe Asn Cys Thr Tyr Ser Asn Ser Ala Ser Gln Ser
20 25 30
Phe Phe Trp Tyr Arg Gln Asp Cys Arg Lys Glu Pro Lys Leu Leu Met
35 40 45
Ser Val Tyr Ser Ser Gly Asn Glu Asp Gly Arg Phe Thr Ala Gln Leu
50 55 60
Asn Arg Ala Ser Gln Tyr Ile Ser Leu Leu Ile Arg Asp Ser Lys Leu
65 70 75 80
Ser Asp Ser Ala Thr Tyr Leu Cys Val Val Asn Arg Asp Asn Tyr Gly
85 90 95
Gln Asn Phe Val Phe Gly Pro Gly Thr Arg Leu Ser Val Leu Pro Tyr
100 105 110
Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser
115 120 125
Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn
130 135 140
Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr Val
145 150 155 160
Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp
165 170 175
Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile
180 185 190
Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val
195 200 205
Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln
210 215 220
Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly
225 230 235 240
Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 772
<211> 252
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 36 - альфа, цистеин-модифицированный
<400> 772
Arg Lys Glu Val Glu Gln Asp Pro Gly Pro Phe Asn Val Pro Glu Gly
1 5 10 15
Ala Thr Val Ala Phe Asn Cys Thr Tyr Ser Asn Ser Ala Ser Gln Ser
20 25 30
Phe Phe Trp Tyr Arg Gln Asp Cys Arg Lys Glu Pro Lys Leu Leu Met
35 40 45
Ser Val Tyr Ser Ser Gly Asn Glu Asp Gly Arg Phe Thr Ala Gln Leu
50 55 60
Asn Arg Ala Ser Gln Tyr Ile Ser Leu Leu Ile Arg Asp Ser Lys Leu
65 70 75 80
Ser Asp Ser Ala Thr Tyr Leu Cys Val Val Asn Arg Asp Asn Tyr Gly
85 90 95
Gln Asn Phe Val Phe Gly Pro Gly Thr Arg Leu Ser Val Leu Pro Tyr
100 105 110
Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser
115 120 125
Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn
130 135 140
Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys Val
145 150 155 160
Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp
165 170 175
Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile
180 185 190
Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val
195 200 205
Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln
210 215 220
Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly
225 230 235 240
Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 773
<211> 274
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 36 - альфа нативный
<400> 773
Met Met Ile Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu
1 5 10 15
Ser Trp Val Trp Ser Gln Arg Lys Glu Val Glu Gln Asp Pro Gly Pro
20 25 30
Phe Asn Val Pro Glu Gly Ala Thr Val Ala Phe Asn Cys Thr Tyr Ser
35 40 45
Asn Ser Ala Ser Gln Ser Phe Phe Trp Tyr Arg Gln Asp Cys Arg Lys
50 55 60
Glu Pro Lys Leu Leu Met Ser Val Tyr Ser Ser Gly Asn Glu Asp Gly
65 70 75 80
Arg Phe Thr Ala Gln Leu Asn Arg Ala Ser Gln Tyr Ile Ser Leu Leu
85 90 95
Ile Arg Asp Ser Lys Leu Ser Asp Ser Ala Thr Tyr Leu Cys Val Val
100 105 110
Asn Arg Asp Asn Tyr Gly Gln Asn Phe Val Phe Gly Pro Gly Thr Arg
115 120 125
Leu Ser Val Leu Pro Tyr Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln
130 135 140
Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp
145 150 155 160
Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr
165 170 175
Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser
180 185 190
Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn
195 200 205
Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro
210 215 220
Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp
225 230 235 240
Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu
245 250 255
Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp
260 265 270
Ser Ser
<210> 774
<211> 274
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 36 - альфа, цистеин-модифицированный
<400> 774
Met Met Ile Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu
1 5 10 15
Ser Trp Val Trp Ser Gln Arg Lys Glu Val Glu Gln Asp Pro Gly Pro
20 25 30
Phe Asn Val Pro Glu Gly Ala Thr Val Ala Phe Asn Cys Thr Tyr Ser
35 40 45
Asn Ser Ala Ser Gln Ser Phe Phe Trp Tyr Arg Gln Asp Cys Arg Lys
50 55 60
Glu Pro Lys Leu Leu Met Ser Val Tyr Ser Ser Gly Asn Glu Asp Gly
65 70 75 80
Arg Phe Thr Ala Gln Leu Asn Arg Ala Ser Gln Tyr Ile Ser Leu Leu
85 90 95
Ile Arg Asp Ser Lys Leu Ser Asp Ser Ala Thr Tyr Leu Cys Val Val
100 105 110
Asn Arg Asp Asn Tyr Gly Gln Asn Phe Val Phe Gly Pro Gly Thr Arg
115 120 125
Leu Ser Val Leu Pro Tyr Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln
130 135 140
Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp
145 150 155 160
Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr
165 170 175
Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser
180 185 190
Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn
195 200 205
Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro
210 215 220
Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp
225 230 235 240
Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu
245 250 255
Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp
260 265 270
Ser Ser
<210> 775
<211> 111
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 36 альфа, вариабельная область
<400> 775
Arg Lys Glu Val Glu Gln Asp Pro Gly Pro Phe Asn Val Pro Glu Gly
1 5 10 15
Ala Thr Val Ala Phe Asn Cys Thr Tyr Ser Asn Ser Ala Ser Gln Ser
20 25 30
Phe Phe Trp Tyr Arg Gln Asp Cys Arg Lys Glu Pro Lys Leu Leu Met
35 40 45
Ser Val Tyr Ser Ser Gly Asn Glu Asp Gly Arg Phe Thr Ala Gln Leu
50 55 60
Asn Arg Ala Ser Gln Tyr Ile Ser Leu Leu Ile Arg Asp Ser Lys Leu
65 70 75 80
Ser Asp Ser Ala Thr Tyr Leu Cys Val Val Asn Arg Asp Asn Tyr Gly
85 90 95
Gln Asn Phe Val Phe Gly Pro Gly Thr Arg Leu Ser Val Leu Pro
100 105 110
<210> 776
<211> 12
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 36 альфа CDR3
<400> 776
Val Val Asn Arg Asp Asn Tyr Gly Gln Asn Phe Val
1 5 10
<210> 777
<211> 292
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 36 - бета нативный
<400> 777
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Met Thr Gly
85 90 95
Arg Ser Asn Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu
115 120 125
Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys
130 135 140
Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val
145 150 155 160
Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro Leu
165 170 175
Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg
180 185 190
Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg
195 200 205
Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln
210 215 220
Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly
225 230 235 240
Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu
245 250 255
Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr
260 265 270
Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys
275 280 285
Asp Ser Arg Gly
290
<210> 778
<211> 292
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 36 - бета, цистеин-модифицированный
<400> 778
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Met Thr Gly
85 90 95
Arg Ser Asn Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu
115 120 125
Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys
130 135 140
Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val
145 150 155 160
Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro Leu
165 170 175
Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg
180 185 190
Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg
195 200 205
Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln
210 215 220
Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly
225 230 235 240
Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu
245 250 255
Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr
260 265 270
Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys
275 280 285
Asp Ser Arg Gly
290
<210> 779
<211> 311
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 36 - бета нативный
<400> 779
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Met Thr Gly Arg Ser Asn Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 780
<211> 311
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 36 - бета, цистеин-модифицированный
<400> 780
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Met Thr Gly Arg Ser Asn Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 781
<211> 113
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 36 бета, вариабельная область
<400> 781
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Met Thr Gly
85 90 95
Arg Ser Asn Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Thr
<210> 782
<211> 11
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 36 бета CDR3
<400> 782
Ala Met Thr Gly Arg Ser Asn Tyr Glu Gln Tyr
1 5 10
<210> 783
<211> 253
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 37 - альфа нативный
<400> 783
Glu Leu Lys Val Glu Gln Asn Pro Leu Phe Leu Ser Met Gln Glu Gly
1 5 10 15
Lys Asn Tyr Thr Ile Tyr Cys Asn Tyr Ser Thr Thr Ser Asp Arg Leu
20 25 30
Tyr Trp Tyr Arg Gln Asp Pro Gly Lys Ser Leu Glu Ser Leu Phe Val
35 40 45
Leu Leu Ser Asn Gly Ala Val Lys Gln Glu Gly Arg Leu Met Ala Ser
50 55 60
Leu Asp Thr Lys Ala Arg Leu Ser Thr Leu His Ile Thr Ala Ala Val
65 70 75 80
His Asp Leu Ser Ala Thr Tyr Phe Cys Ala Gly Tyr Ser Gly Ala Gly
85 90 95
Ser Tyr Gln Leu Thr Phe Gly Lys Gly Thr Lys Leu Ser Val Ile Pro
100 105 110
Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys
115 120 125
Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr
130 135 140
Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr
145 150 155 160
Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala
165 170 175
Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser
180 185 190
Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp
195 200 205
Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe
210 215 220
Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala
225 230 235 240
Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 784
<211> 253
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 37 - альфа, цистеин-модифицированный
<400> 784
Glu Leu Lys Val Glu Gln Asn Pro Leu Phe Leu Ser Met Gln Glu Gly
1 5 10 15
Lys Asn Tyr Thr Ile Tyr Cys Asn Tyr Ser Thr Thr Ser Asp Arg Leu
20 25 30
Tyr Trp Tyr Arg Gln Asp Pro Gly Lys Ser Leu Glu Ser Leu Phe Val
35 40 45
Leu Leu Ser Asn Gly Ala Val Lys Gln Glu Gly Arg Leu Met Ala Ser
50 55 60
Leu Asp Thr Lys Ala Arg Leu Ser Thr Leu His Ile Thr Ala Ala Val
65 70 75 80
His Asp Leu Ser Ala Thr Tyr Phe Cys Ala Gly Tyr Ser Gly Ala Gly
85 90 95
Ser Tyr Gln Leu Thr Phe Gly Lys Gly Thr Lys Leu Ser Val Ile Pro
100 105 110
Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys
115 120 125
Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr
130 135 140
Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys
145 150 155 160
Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala
165 170 175
Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser
180 185 190
Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp
195 200 205
Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe
210 215 220
Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala
225 230 235 240
Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 785
<211> 271
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 37 - альфа нативный
<400> 785
Met Lys Lys Leu Leu Ala Met Ile Leu Trp Leu Gln Leu Asp Arg Leu
1 5 10 15
Ser Gly Glu Leu Lys Val Glu Gln Asn Pro Leu Phe Leu Ser Met Gln
20 25 30
Glu Gly Lys Asn Tyr Thr Ile Tyr Cys Asn Tyr Ser Thr Thr Ser Asp
35 40 45
Arg Leu Tyr Trp Tyr Arg Gln Asp Pro Gly Lys Ser Leu Glu Ser Leu
50 55 60
Phe Val Leu Leu Ser Asn Gly Ala Val Lys Gln Glu Gly Arg Leu Met
65 70 75 80
Ala Ser Leu Asp Thr Lys Ala Arg Leu Ser Thr Leu His Ile Thr Ala
85 90 95
Ala Val His Asp Leu Ser Ala Thr Tyr Phe Cys Ala Gly Tyr Ser Gly
100 105 110
Ala Gly Ser Tyr Gln Leu Thr Phe Gly Lys Gly Thr Lys Leu Ser Val
115 120 125
Ile Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp
130 135 140
Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser
145 150 155 160
Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp
165 170 175
Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala
180 185 190
Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn
195 200 205
Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser
210 215 220
Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu
225 230 235 240
Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys
245 250 255
Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
260 265 270
<210> 786
<211> 271
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 37 - альфа, цистеин-модифицированный
<400> 786
Met Lys Lys Leu Leu Ala Met Ile Leu Trp Leu Gln Leu Asp Arg Leu
1 5 10 15
Ser Gly Glu Leu Lys Val Glu Gln Asn Pro Leu Phe Leu Ser Met Gln
20 25 30
Glu Gly Lys Asn Tyr Thr Ile Tyr Cys Asn Tyr Ser Thr Thr Ser Asp
35 40 45
Arg Leu Tyr Trp Tyr Arg Gln Asp Pro Gly Lys Ser Leu Glu Ser Leu
50 55 60
Phe Val Leu Leu Ser Asn Gly Ala Val Lys Gln Glu Gly Arg Leu Met
65 70 75 80
Ala Ser Leu Asp Thr Lys Ala Arg Leu Ser Thr Leu His Ile Thr Ala
85 90 95
Ala Val His Asp Leu Ser Ala Thr Tyr Phe Cys Ala Gly Tyr Ser Gly
100 105 110
Ala Gly Ser Tyr Gln Leu Thr Phe Gly Lys Gly Thr Lys Leu Ser Val
115 120 125
Ile Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp
130 135 140
Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser
145 150 155 160
Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp
165 170 175
Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala
180 185 190
Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn
195 200 205
Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser
210 215 220
Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu
225 230 235 240
Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys
245 250 255
Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
260 265 270
<210> 787
<211> 112
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 37 альфа, вариабельная область
<400> 787
Glu Leu Lys Val Glu Gln Asn Pro Leu Phe Leu Ser Met Gln Glu Gly
1 5 10 15
Lys Asn Tyr Thr Ile Tyr Cys Asn Tyr Ser Thr Thr Ser Asp Arg Leu
20 25 30
Tyr Trp Tyr Arg Gln Asp Pro Gly Lys Ser Leu Glu Ser Leu Phe Val
35 40 45
Leu Leu Ser Asn Gly Ala Val Lys Gln Glu Gly Arg Leu Met Ala Ser
50 55 60
Leu Asp Thr Lys Ala Arg Leu Ser Thr Leu His Ile Thr Ala Ala Val
65 70 75 80
His Asp Leu Ser Ala Thr Tyr Phe Cys Ala Gly Tyr Ser Gly Ala Gly
85 90 95
Ser Tyr Gln Leu Thr Phe Gly Lys Gly Thr Lys Leu Ser Val Ile Pro
100 105 110
<210> 788
<211> 12
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 37 альфа CDR3
<400> 788
Ala Gly Tyr Ser Gly Ala Gly Ser Tyr Gln Leu Thr
1 5 10
<210> 789
<211> 291
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 37 - бета нативный
<400> 789
Gly Ala Gly Val Ser Gln Ser Pro Arg Tyr Lys Val Ala Lys Arg Gly
1 5 10 15
Gln Asp Val Ala Leu Arg Cys Asp Pro Ile Ser Gly His Val Ser Leu
20 25 30
Phe Trp Tyr Gln Gln Ala Leu Gly Gln Gly Pro Glu Phe Leu Thr Tyr
35 40 45
Phe Gln Asn Glu Ala Gln Leu Asp Lys Ser Gly Leu Pro Ser Asp Arg
50 55 60
Phe Phe Ala Glu Arg Pro Glu Gly Ser Val Ser Thr Leu Lys Ile Gln
65 70 75 80
Arg Thr Gln Gln Glu Asp Ser Ala Val Tyr Leu Cys Ala Ser Ser Leu
85 90 95
Leu Leu Gly Ala Tyr Gly Tyr Thr Phe Gly Ser Gly Thr Arg Leu Thr
100 105 110
Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val Phe
115 120 125
Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val
130 135 140
Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp Trp
145 150 155 160
Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro
165 170 175
Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser
180 185 190
Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe
195 200 205
Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr
210 215 220
Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp
225 230 235 240
Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly Val
245 250 255
Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu
260 265 270
Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg
275 280 285
Lys Asp Phe
290
<210> 790
<211> 291
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 37 - бета
цистеин-модифицированный
<400> 790
Gly Ala Gly Val Ser Gln Ser Pro Arg Tyr Lys Val Ala Lys Arg Gly
1 5 10 15
Gln Asp Val Ala Leu Arg Cys Asp Pro Ile Ser Gly His Val Ser Leu
20 25 30
Phe Trp Tyr Gln Gln Ala Leu Gly Gln Gly Pro Glu Phe Leu Thr Tyr
35 40 45
Phe Gln Asn Glu Ala Gln Leu Asp Lys Ser Gly Leu Pro Ser Asp Arg
50 55 60
Phe Phe Ala Glu Arg Pro Glu Gly Ser Val Ser Thr Leu Lys Ile Gln
65 70 75 80
Arg Thr Gln Gln Glu Asp Ser Ala Val Tyr Leu Cys Ala Ser Ser Leu
85 90 95
Leu Leu Gly Ala Tyr Gly Tyr Thr Phe Gly Ser Gly Thr Arg Leu Thr
100 105 110
Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val Phe
115 120 125
Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val
130 135 140
Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp Trp
145 150 155 160
Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro
165 170 175
Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser
180 185 190
Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe
195 200 205
Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr
210 215 220
Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp
225 230 235 240
Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly Val
245 250 255
Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu
260 265 270
Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg
275 280 285
Lys Asp Phe
290
<210> 791
<211> 310
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 37 - бета нативный
<400> 791
Met Gly Thr Arg Leu Leu Cys Trp Val Val Leu Gly Phe Leu Gly Thr
1 5 10 15
Asp His Thr Gly Ala Gly Val Ser Gln Ser Pro Arg Tyr Lys Val Ala
20 25 30
Lys Arg Gly Gln Asp Val Ala Leu Arg Cys Asp Pro Ile Ser Gly His
35 40 45
Val Ser Leu Phe Trp Tyr Gln Gln Ala Leu Gly Gln Gly Pro Glu Phe
50 55 60
Leu Thr Tyr Phe Gln Asn Glu Ala Gln Leu Asp Lys Ser Gly Leu Pro
65 70 75 80
Ser Asp Arg Phe Phe Ala Glu Arg Pro Glu Gly Ser Val Ser Thr Leu
85 90 95
Lys Ile Gln Arg Thr Gln Gln Glu Asp Ser Ala Val Tyr Leu Cys Ala
100 105 110
Ser Ser Leu Leu Leu Gly Ala Tyr Gly Tyr Thr Phe Gly Ser Gly Thr
115 120 125
Arg Leu Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe
305 310
<210> 792
<211> 310
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 37 - бета, цистеин-модифицированный
<400> 792
Met Gly Thr Arg Leu Leu Cys Trp Val Val Leu Gly Phe Leu Gly Thr
1 5 10 15
Asp His Thr Gly Ala Gly Val Ser Gln Ser Pro Arg Tyr Lys Val Ala
20 25 30
Lys Arg Gly Gln Asp Val Ala Leu Arg Cys Asp Pro Ile Ser Gly His
35 40 45
Val Ser Leu Phe Trp Tyr Gln Gln Ala Leu Gly Gln Gly Pro Glu Phe
50 55 60
Leu Thr Tyr Phe Gln Asn Glu Ala Gln Leu Asp Lys Ser Gly Leu Pro
65 70 75 80
Ser Asp Arg Phe Phe Ala Glu Arg Pro Glu Gly Ser Val Ser Thr Leu
85 90 95
Lys Ile Gln Arg Thr Gln Gln Glu Asp Ser Ala Val Tyr Leu Cys Ala
100 105 110
Ser Ser Leu Leu Leu Gly Ala Tyr Gly Tyr Thr Phe Gly Ser Gly Thr
115 120 125
Arg Leu Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe
305 310
<210> 793
<211> 114
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 37 бета, вариабельная область
<400> 793
Gly Ala Gly Val Ser Gln Ser Pro Arg Tyr Lys Val Ala Lys Arg Gly
1 5 10 15
Gln Asp Val Ala Leu Arg Cys Asp Pro Ile Ser Gly His Val Ser Leu
20 25 30
Phe Trp Tyr Gln Gln Ala Leu Gly Gln Gly Pro Glu Phe Leu Thr Tyr
35 40 45
Phe Gln Asn Glu Ala Gln Leu Asp Lys Ser Gly Leu Pro Ser Asp Arg
50 55 60
Phe Phe Ala Glu Arg Pro Glu Gly Ser Val Ser Thr Leu Lys Ile Gln
65 70 75 80
Arg Thr Gln Gln Glu Asp Ser Ala Val Tyr Leu Cys Ala Ser Ser Leu
85 90 95
Leu Leu Gly Ala Tyr Gly Tyr Thr Phe Gly Ser Gly Thr Arg Leu Thr
100 105 110
Val Val
<210> 794
<211> 12
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 37 бета CDR3
<400> 794
Ala Ser Ser Leu Leu Leu Gly Ala Tyr Gly Tyr Thr
1 5 10
<210> 795
<211> 247
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 38 - альфа нативный
<400> 795
Gly Ile Gln Val Glu Gln Ser Pro Pro Asp Leu Ile Leu Gln Glu Gly
1 5 10 15
Ala Asn Ser Thr Leu Arg Cys Asn Phe Ser Asp Ser Val Asn Asn Leu
20 25 30
Gln Trp Phe His Gln Asn Pro Trp Gly Gln Leu Ile Asn Leu Phe Tyr
35 40 45
Ile Pro Ser Gly Thr Lys Gln Asn Gly Arg Leu Ser Ala Thr Thr Val
50 55 60
Ala Thr Glu Arg Tyr Ser Leu Leu Tyr Ile Ser Ser Ser Gln Thr Thr
65 70 75 80
Asp Ser Gly Val Tyr Phe Cys Ala Val Gly Phe Asn Asp Met Arg Phe
85 90 95
Gly Ala Gly Thr Arg Leu Thr Val Lys Pro Asn Ile Gln Asn Pro Asp
100 105 110
Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val
115 120 125
Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys
130 135 140
Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser
145 150 155 160
Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp
165 170 175
Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr
180 185 190
Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys
195 200 205
Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile
210 215 220
Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met
225 230 235 240
Thr Leu Arg Leu Trp Ser Ser
245
<210> 796
<211> 247
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 38 - альфа, цистеин-модифицированный
<400> 796
Gly Ile Gln Val Glu Gln Ser Pro Pro Asp Leu Ile Leu Gln Glu Gly
1 5 10 15
Ala Asn Ser Thr Leu Arg Cys Asn Phe Ser Asp Ser Val Asn Asn Leu
20 25 30
Gln Trp Phe His Gln Asn Pro Trp Gly Gln Leu Ile Asn Leu Phe Tyr
35 40 45
Ile Pro Ser Gly Thr Lys Gln Asn Gly Arg Leu Ser Ala Thr Thr Val
50 55 60
Ala Thr Glu Arg Tyr Ser Leu Leu Tyr Ile Ser Ser Ser Gln Thr Thr
65 70 75 80
Asp Ser Gly Val Tyr Phe Cys Ala Val Gly Phe Asn Asp Met Arg Phe
85 90 95
Gly Ala Gly Thr Arg Leu Thr Val Lys Pro Asn Ile Gln Asn Pro Asp
100 105 110
Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val
115 120 125
Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys
130 135 140
Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser
145 150 155 160
Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp
165 170 175
Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr
180 185 190
Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys
195 200 205
Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile
210 215 220
Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met
225 230 235 240
Thr Leu Arg Leu Trp Ser Ser
245
<210> 797
<211> 279
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 38 - альфа нативный
<400> 797
Met Val Lys Met Pro Gly Ala Arg Arg Gln Ser Ile Met Lys Arg Ile
1 5 10 15
Leu Gly Ala Leu Leu Gly Leu Leu Ser Ala Gln Val Cys Cys Val Arg
20 25 30
Gly Ile Gln Val Glu Gln Ser Pro Pro Asp Leu Ile Leu Gln Glu Gly
35 40 45
Ala Asn Ser Thr Leu Arg Cys Asn Phe Ser Asp Ser Val Asn Asn Leu
50 55 60
Gln Trp Phe His Gln Asn Pro Trp Gly Gln Leu Ile Asn Leu Phe Tyr
65 70 75 80
Ile Pro Ser Gly Thr Lys Gln Asn Gly Arg Leu Ser Ala Thr Thr Val
85 90 95
Ala Thr Glu Arg Tyr Ser Leu Leu Tyr Ile Ser Ser Ser Gln Thr Thr
100 105 110
Asp Ser Gly Val Tyr Phe Cys Ala Val Gly Phe Asn Asp Met Arg Phe
115 120 125
Gly Ala Gly Thr Arg Leu Thr Val Lys Pro Asn Ile Gln Asn Pro Asp
130 135 140
Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val
145 150 155 160
Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys
165 170 175
Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser
180 185 190
Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp
195 200 205
Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr
210 215 220
Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys
225 230 235 240
Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile
245 250 255
Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met
260 265 270
Thr Leu Arg Leu Trp Ser Ser
275
<210> 798
<211> 279
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 38 - альфа, цистеин-модифицированный
<400> 798
Met Val Lys Met Pro Gly Ala Arg Arg Gln Ser Ile Met Lys Arg Ile
1 5 10 15
Leu Gly Ala Leu Leu Gly Leu Leu Ser Ala Gln Val Cys Cys Val Arg
20 25 30
Gly Ile Gln Val Glu Gln Ser Pro Pro Asp Leu Ile Leu Gln Glu Gly
35 40 45
Ala Asn Ser Thr Leu Arg Cys Asn Phe Ser Asp Ser Val Asn Asn Leu
50 55 60
Gln Trp Phe His Gln Asn Pro Trp Gly Gln Leu Ile Asn Leu Phe Tyr
65 70 75 80
Ile Pro Ser Gly Thr Lys Gln Asn Gly Arg Leu Ser Ala Thr Thr Val
85 90 95
Ala Thr Glu Arg Tyr Ser Leu Leu Tyr Ile Ser Ser Ser Gln Thr Thr
100 105 110
Asp Ser Gly Val Tyr Phe Cys Ala Val Gly Phe Asn Asp Met Arg Phe
115 120 125
Gly Ala Gly Thr Arg Leu Thr Val Lys Pro Asn Ile Gln Asn Pro Asp
130 135 140
Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val
145 150 155 160
Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys
165 170 175
Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser
180 185 190
Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp
195 200 205
Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr
210 215 220
Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys
225 230 235 240
Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile
245 250 255
Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met
260 265 270
Thr Leu Arg Leu Trp Ser Ser
275
<210> 799
<211> 106
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 38 альфа, вариабельная область
<400> 799
Gly Ile Gln Val Glu Gln Ser Pro Pro Asp Leu Ile Leu Gln Glu Gly
1 5 10 15
Ala Asn Ser Thr Leu Arg Cys Asn Phe Ser Asp Ser Val Asn Asn Leu
20 25 30
Gln Trp Phe His Gln Asn Pro Trp Gly Gln Leu Ile Asn Leu Phe Tyr
35 40 45
Ile Pro Ser Gly Thr Lys Gln Asn Gly Arg Leu Ser Ala Thr Thr Val
50 55 60
Ala Thr Glu Arg Tyr Ser Leu Leu Tyr Ile Ser Ser Ser Gln Thr Thr
65 70 75 80
Asp Ser Gly Val Tyr Phe Cys Ala Val Gly Phe Asn Asp Met Arg Phe
85 90 95
Gly Ala Gly Thr Arg Leu Thr Val Lys Pro
100 105
<210> 800
<211> 5
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 38 альфа CDR1
<400> 800
Asp Ser Val Asn Asn
1 5
<210> 801
<211> 5
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 38 альфа CDR2
<400> 801
Ile Pro Ser Gly Thr
1 5
<210> 802
<211> 8
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 38 альфа CDR3
<400> 802
Ala Val Gly Phe Asn Asp Met Arg
1 5
<210> 803
<211> 32
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 38 альфа, Сигнальный пептид
<400> 803
Met Val Lys Met Pro Gly Ala Arg Arg Gln Ser Ile Met Lys Arg Ile
1 5 10 15
Leu Gly Ala Leu Leu Gly Leu Leu Ser Ala Gln Val Cys Cys Val Arg
20 25 30
<210> 804
<211> 292
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 38 - бета нативный
<400> 804
Glu Thr Gly Val Thr Gln Ser Pro Thr His Leu Ile Lys Thr Arg Gly
1 5 10 15
Gln Gln Val Thr Leu Arg Cys Ser Ser Gln Ser Gly His Asn Thr Val
20 25 30
Ser Trp Tyr Gln Gln Ala Leu Gly Gln Gly Pro Gln Phe Ile Phe Gln
35 40 45
Tyr Tyr Arg Glu Glu Glu Asn Gly Arg Gly Asn Phe Pro Pro Arg Phe
50 55 60
Ser Gly Leu Gln Phe Pro Asn Tyr Ser Ser Glu Leu Asn Val Asn Ala
65 70 75 80
Leu Glu Leu Asp Asp Ser Ala Leu Tyr Leu Cys Ala Ser Ser Leu Val
85 90 95
Ala Gly Gly Glu Thr Gln Tyr Phe Gly Pro Gly Thr Arg Leu Leu Val
100 105 110
Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu
115 120 125
Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys
130 135 140
Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val
145 150 155 160
Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro Leu
165 170 175
Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg
180 185 190
Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg
195 200 205
Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln
210 215 220
Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly
225 230 235 240
Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu
245 250 255
Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr
260 265 270
Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys
275 280 285
Asp Ser Arg Gly
290
<210> 805
<211> 292
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 38 - бета, цистеин-модифицированный
<400> 805
Glu Thr Gly Val Thr Gln Ser Pro Thr His Leu Ile Lys Thr Arg Gly
1 5 10 15
Gln Gln Val Thr Leu Arg Cys Ser Ser Gln Ser Gly His Asn Thr Val
20 25 30
Ser Trp Tyr Gln Gln Ala Leu Gly Gln Gly Pro Gln Phe Ile Phe Gln
35 40 45
Tyr Tyr Arg Glu Glu Glu Asn Gly Arg Gly Asn Phe Pro Pro Arg Phe
50 55 60
Ser Gly Leu Gln Phe Pro Asn Tyr Ser Ser Glu Leu Asn Val Asn Ala
65 70 75 80
Leu Glu Leu Asp Asp Ser Ala Leu Tyr Leu Cys Ala Ser Ser Leu Val
85 90 95
Ala Gly Gly Glu Thr Gln Tyr Phe Gly Pro Gly Thr Arg Leu Leu Val
100 105 110
Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu
115 120 125
Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys
130 135 140
Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val
145 150 155 160
Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro Leu
165 170 175
Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg
180 185 190
Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg
195 200 205
Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln
210 215 220
Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly
225 230 235 240
Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu
245 250 255
Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr
260 265 270
Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys
275 280 285
Asp Ser Arg Gly
290
<210> 806
<211> 311
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 38 - бета нативный
<400> 806
Met Gly Pro Gly Leu Leu Cys Trp Val Leu Leu Cys Leu Leu Gly Ala
1 5 10 15
Gly Ser Val Glu Thr Gly Val Thr Gln Ser Pro Thr His Leu Ile Lys
20 25 30
Thr Arg Gly Gln Gln Val Thr Leu Arg Cys Ser Ser Gln Ser Gly His
35 40 45
Asn Thr Val Ser Trp Tyr Gln Gln Ala Leu Gly Gln Gly Pro Gln Phe
50 55 60
Ile Phe Gln Tyr Tyr Arg Glu Glu Glu Asn Gly Arg Gly Asn Phe Pro
65 70 75 80
Pro Arg Phe Ser Gly Leu Gln Phe Pro Asn Tyr Ser Ser Glu Leu Asn
85 90 95
Val Asn Ala Leu Glu Leu Asp Asp Ser Ala Leu Tyr Leu Cys Ala Ser
100 105 110
Ser Leu Val Ala Gly Gly Glu Thr Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Leu Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 807
<211> 311
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 38 - бета, цистеин-модифицированный
<400> 807
Met Gly Pro Gly Leu Leu Cys Trp Val Leu Leu Cys Leu Leu Gly Ala
1 5 10 15
Gly Ser Val Glu Thr Gly Val Thr Gln Ser Pro Thr His Leu Ile Lys
20 25 30
Thr Arg Gly Gln Gln Val Thr Leu Arg Cys Ser Ser Gln Ser Gly His
35 40 45
Asn Thr Val Ser Trp Tyr Gln Gln Ala Leu Gly Gln Gly Pro Gln Phe
50 55 60
Ile Phe Gln Tyr Tyr Arg Glu Glu Glu Asn Gly Arg Gly Asn Phe Pro
65 70 75 80
Pro Arg Phe Ser Gly Leu Gln Phe Pro Asn Tyr Ser Ser Glu Leu Asn
85 90 95
Val Asn Ala Leu Glu Leu Asp Asp Ser Ala Leu Tyr Leu Cys Ala Ser
100 105 110
Ser Leu Val Ala Gly Gly Glu Thr Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Leu Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 808
<211> 113
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 38 бета, вариабельная область
<400> 808
Glu Thr Gly Val Thr Gln Ser Pro Thr His Leu Ile Lys Thr Arg Gly
1 5 10 15
Gln Gln Val Thr Leu Arg Cys Ser Ser Gln Ser Gly His Asn Thr Val
20 25 30
Ser Trp Tyr Gln Gln Ala Leu Gly Gln Gly Pro Gln Phe Ile Phe Gln
35 40 45
Tyr Tyr Arg Glu Glu Glu Asn Gly Arg Gly Asn Phe Pro Pro Arg Phe
50 55 60
Ser Gly Leu Gln Phe Pro Asn Tyr Ser Ser Glu Leu Asn Val Asn Ala
65 70 75 80
Leu Glu Leu Asp Asp Ser Ala Leu Tyr Leu Cys Ala Ser Ser Leu Val
85 90 95
Ala Gly Gly Glu Thr Gln Tyr Phe Gly Pro Gly Thr Arg Leu Leu Val
100 105 110
Leu
<210> 809
<211> 12
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 38 бета CDR3
<400> 809
Ala Ser Ser Leu Val Ala Gly Gly Glu Thr Gln Tyr
1 5 10
<210> 810
<211> 19
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 38 бета, Сигнальный пептид
<400> 810
Met Gly Pro Gly Leu Leu Cys Trp Val Leu Leu Cys Leu Leu Gly Ala
1 5 10 15
Gly Ser Val
<210> 811
<211> 253
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 39 - альфа нативный
<400> 811
Gly Gln Gln Leu Asn Gln Ser Pro Gln Ser Met Phe Ile Gln Glu Gly
1 5 10 15
Glu Asp Val Ser Met Asn Cys Thr Ser Ser Ser Ile Phe Asn Thr Trp
20 25 30
Leu Trp Tyr Lys Gln Glu Pro Gly Glu Gly Pro Val Leu Leu Ile Ala
35 40 45
Leu Tyr Lys Ala Gly Glu Leu Thr Ser Asn Gly Arg Leu Thr Ala Gln
50 55 60
Phe Gly Ile Thr Arg Lys Asp Ser Phe Leu Asn Ile Ser Ala Ser Ile
65 70 75 80
Pro Ser Asp Val Gly Ile Tyr Phe Cys Ala Gly Tyr Ser Ser Ser Asn
85 90 95
Asp Tyr Lys Leu Ser Phe Gly Ala Gly Thr Thr Val Thr Val Arg Ala
100 105 110
Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys
115 120 125
Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr
130 135 140
Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr
145 150 155 160
Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala
165 170 175
Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser
180 185 190
Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp
195 200 205
Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe
210 215 220
Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala
225 230 235 240
Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 812
<211> 253
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 39 - альфа, цистеин-модифицированный
<400> 812
Gly Gln Gln Leu Asn Gln Ser Pro Gln Ser Met Phe Ile Gln Glu Gly
1 5 10 15
Glu Asp Val Ser Met Asn Cys Thr Ser Ser Ser Ile Phe Asn Thr Trp
20 25 30
Leu Trp Tyr Lys Gln Glu Pro Gly Glu Gly Pro Val Leu Leu Ile Ala
35 40 45
Leu Tyr Lys Ala Gly Glu Leu Thr Ser Asn Gly Arg Leu Thr Ala Gln
50 55 60
Phe Gly Ile Thr Arg Lys Asp Ser Phe Leu Asn Ile Ser Ala Ser Ile
65 70 75 80
Pro Ser Asp Val Gly Ile Tyr Phe Cys Ala Gly Tyr Ser Ser Ser Asn
85 90 95
Asp Tyr Lys Leu Ser Phe Gly Ala Gly Thr Thr Val Thr Val Arg Ala
100 105 110
Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys
115 120 125
Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr
130 135 140
Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys
145 150 155 160
Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala
165 170 175
Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser
180 185 190
Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp
195 200 205
Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe
210 215 220
Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala
225 230 235 240
Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 813
<211> 260
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 39 - альфа нативный
<400> 813
Met Gln Leu Thr Trp Val Ser Gly Gln Gln Leu Asn Gln Ser Pro Gln
1 5 10 15
Ser Met Phe Ile Gln Glu Gly Glu Asp Val Ser Met Asn Cys Thr Ser
20 25 30
Ser Ser Ile Phe Asn Thr Trp Leu Trp Tyr Lys Gln Glu Pro Gly Glu
35 40 45
Gly Pro Val Leu Leu Ile Ala Leu Tyr Lys Ala Gly Glu Leu Thr Ser
50 55 60
Asn Gly Arg Leu Thr Ala Gln Phe Gly Ile Thr Arg Lys Asp Ser Phe
65 70 75 80
Leu Asn Ile Ser Ala Ser Ile Pro Ser Asp Val Gly Ile Tyr Phe Cys
85 90 95
Ala Gly Tyr Ser Ser Ser Asn Asp Tyr Lys Leu Ser Phe Gly Ala Gly
100 105 110
Thr Thr Val Thr Val Arg Ala Asn Ile Gln Asn Pro Asp Pro Ala Val
115 120 125
Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe
130 135 140
Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp
145 150 155 160
Val Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe
165 170 175
Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys
180 185 190
Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro
195 200 205
Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu
210 215 220
Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg
225 230 235 240
Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg
245 250 255
Leu Trp Ser Ser
260
<210> 814
<211> 260
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 39 - альфа
цистеин-модифицированный
<400> 814
Met Gln Leu Thr Trp Val Ser Gly Gln Gln Leu Asn Gln Ser Pro Gln
1 5 10 15
Ser Met Phe Ile Gln Glu Gly Glu Asp Val Ser Met Asn Cys Thr Ser
20 25 30
Ser Ser Ile Phe Asn Thr Trp Leu Trp Tyr Lys Gln Glu Pro Gly Glu
35 40 45
Gly Pro Val Leu Leu Ile Ala Leu Tyr Lys Ala Gly Glu Leu Thr Ser
50 55 60
Asn Gly Arg Leu Thr Ala Gln Phe Gly Ile Thr Arg Lys Asp Ser Phe
65 70 75 80
Leu Asn Ile Ser Ala Ser Ile Pro Ser Asp Val Gly Ile Tyr Phe Cys
85 90 95
Ala Gly Tyr Ser Ser Ser Asn Asp Tyr Lys Leu Ser Phe Gly Ala Gly
100 105 110
Thr Thr Val Thr Val Arg Ala Asn Ile Gln Asn Pro Asp Pro Ala Val
115 120 125
Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe
130 135 140
Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp
145 150 155 160
Val Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe
165 170 175
Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys
180 185 190
Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro
195 200 205
Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu
210 215 220
Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg
225 230 235 240
Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg
245 250 255
Leu Trp Ser Ser
260
<210> 815
<211> 112
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 39 альфа, вариабельная область
<400> 815
Gly Gln Gln Leu Asn Gln Ser Pro Gln Ser Met Phe Ile Gln Glu Gly
1 5 10 15
Glu Asp Val Ser Met Asn Cys Thr Ser Ser Ser Ile Phe Asn Thr Trp
20 25 30
Leu Trp Tyr Lys Gln Glu Pro Gly Glu Gly Pro Val Leu Leu Ile Ala
35 40 45
Leu Tyr Lys Ala Gly Glu Leu Thr Ser Asn Gly Arg Leu Thr Ala Gln
50 55 60
Phe Gly Ile Thr Arg Lys Asp Ser Phe Leu Asn Ile Ser Ala Ser Ile
65 70 75 80
Pro Ser Asp Val Gly Ile Tyr Phe Cys Ala Gly Tyr Ser Ser Ser Asn
85 90 95
Asp Tyr Lys Leu Ser Phe Gly Ala Gly Thr Thr Val Thr Val Arg Ala
100 105 110
<210> 816
<211> 5
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 39/ TCR 40/ TCR 42/ TCR 45 альфа CDR1
<400> 816
Thr Thr Leu Ser Asn
1 5
<210> 817
<211> 7
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 39 альфа CDR2
<400> 817
Leu Tyr Lys Ala Gly Glu Leu
1 5
<210> 818
<211> 12
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 39 альфа CDR3
<400> 818
Ala Gly Tyr Ser Ser Ser Asn Asp Tyr Lys Leu Ser
1 5 10
<210> 819
<211> 7
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 39 альфа, Сигнальный пептид
<400> 819
Met Gln Leu Thr Trp Val Ser
1 5
<210> 820
<211> 291
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 39 - бета нативный
<400> 820
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Ser Thr Pro
85 90 95
Arg Asp Arg Gly Lys Glu Ala Phe Phe Gly Gln Gly Thr Arg Leu Thr
100 105 110
Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val Phe
115 120 125
Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val
130 135 140
Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp Trp
145 150 155 160
Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro
165 170 175
Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser
180 185 190
Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe
195 200 205
Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr
210 215 220
Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp
225 230 235 240
Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly Val
245 250 255
Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu
260 265 270
Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg
275 280 285
Lys Asp Phe
290
<210> 821
<211> 291
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 39 - бета
цистеин-модифицированный
<400> 821
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Ser Thr Pro
85 90 95
Arg Asp Arg Gly Lys Glu Ala Phe Phe Gly Gln Gly Thr Arg Leu Thr
100 105 110
Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val Phe
115 120 125
Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val
130 135 140
Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp Trp
145 150 155 160
Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro
165 170 175
Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser
180 185 190
Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe
195 200 205
Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr
210 215 220
Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp
225 230 235 240
Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly Val
245 250 255
Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu
260 265 270
Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg
275 280 285
Lys Asp Phe
290
<210> 822
<211> 310
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 39 - бета нативный
<400> 822
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ser Thr Pro Arg Asp Arg Gly Lys Glu Ala Phe Phe Gly Gln Gly Thr
115 120 125
Arg Leu Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe
305 310
<210> 823
<211> 310
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 39 - бета, цистеин-модифицированный
<400> 823
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ser Thr Pro Arg Asp Arg Gly Lys Glu Ala Phe Phe Gly Gln Gly Thr
115 120 125
Arg Leu Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val
130 135 140
Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala
145 150 155 160
Thr Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu
165 170 175
Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp
180 185 190
Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys
195 200 205
Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg
210 215 220
Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp
225 230 235 240
Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala
245 250 255
Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln
260 265 270
Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys
275 280 285
Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met
290 295 300
Val Lys Arg Lys Asp Phe
305 310
<210> 824
<211> 114
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 39 бета, вариабельная область
<400> 824
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Ser Thr Pro
85 90 95
Arg Asp Arg Gly Lys Glu Ala Phe Phe Gly Gln Gly Thr Arg Leu Thr
100 105 110
Val Val
<210> 825
<211> 12
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 39 бета CDR3
<400> 825
Ala Ser Thr Pro Arg Asp Arg Gly Lys Glu Ala Phe
1 5 10
<210> 826
<211> 251
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 40 - альфа нативный
<400> 826
Gly Gln Gln Val Met Gln Ile Pro Gln Tyr Gln His Val Gln Glu Gly
1 5 10 15
Glu Asp Phe Thr Thr Tyr Cys Asn Ser Ser Thr Thr Leu Ser Asn Ile
20 25 30
Gln Trp Tyr Lys Gln Arg Pro Gly Gly His Pro Val Phe Leu Ile Gln
35 40 45
Leu Val Lys Ser Gly Glu Val Lys Lys Gln Lys Arg Leu Thr Phe Gln
50 55 60
Phe Gly Glu Ala Lys Lys Asn Ser Ser Leu His Ile Thr Ala Thr Gln
65 70 75 80
Thr Thr Asp Val Gly Thr Tyr Phe Cys Ala Gly Arg Asn Asn Phe Asn
85 90 95
Lys Phe Tyr Phe Gly Ser Gly Thr Lys Leu Asn Val Lys Pro Asn Ile
100 105 110
Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser
115 120 125
Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val
130 135 140
Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr Val Leu
145 150 155 160
Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser
165 170 175
Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile
180 185 190
Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys
195 200 205
Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn
210 215 220
Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe
225 230 235 240
Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 827
<211> 251
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 40 - альфа, цистеин-модифицированный
<400> 827
Gly Gln Gln Val Met Gln Ile Pro Gln Tyr Gln His Val Gln Glu Gly
1 5 10 15
Glu Asp Phe Thr Thr Tyr Cys Asn Ser Ser Thr Thr Leu Ser Asn Ile
20 25 30
Gln Trp Tyr Lys Gln Arg Pro Gly Gly His Pro Val Phe Leu Ile Gln
35 40 45
Leu Val Lys Ser Gly Glu Val Lys Lys Gln Lys Arg Leu Thr Phe Gln
50 55 60
Phe Gly Glu Ala Lys Lys Asn Ser Ser Leu His Ile Thr Ala Thr Gln
65 70 75 80
Thr Thr Asp Val Gly Thr Tyr Phe Cys Ala Gly Arg Asn Asn Phe Asn
85 90 95
Lys Phe Tyr Phe Gly Ser Gly Thr Lys Leu Asn Val Lys Pro Asn Ile
100 105 110
Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser
115 120 125
Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val
130 135 140
Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys Val Leu
145 150 155 160
Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser
165 170 175
Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile
180 185 190
Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys
195 200 205
Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn
210 215 220
Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe
225 230 235 240
Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 828
<211> 305
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 40 - альфа нативный
<400> 828
Met His Thr Ser Thr Phe Gln Asn Arg Pro Gln Leu Phe Leu Leu Ile
1 5 10 15
Trp Lys Lys Leu Val Pro Gly Asn Pro Phe Arg Arg Ser Trp Met Lys
20 25 30
Arg Glu Arg Glu Met Leu Leu Ile Thr Ser Met Leu Val Leu Trp Met
35 40 45
Gln Leu Ser Gln Val Asn Gly Gln Gln Val Met Gln Ile Pro Gln Tyr
50 55 60
Gln His Val Gln Glu Gly Glu Asp Phe Thr Thr Tyr Cys Asn Ser Ser
65 70 75 80
Thr Thr Leu Ser Asn Ile Gln Trp Tyr Lys Gln Arg Pro Gly Gly His
85 90 95
Pro Val Phe Leu Ile Gln Leu Val Lys Ser Gly Glu Val Lys Lys Gln
100 105 110
Lys Arg Leu Thr Phe Gln Phe Gly Glu Ala Lys Lys Asn Ser Ser Leu
115 120 125
His Ile Thr Ala Thr Gln Thr Thr Asp Val Gly Thr Tyr Phe Cys Ala
130 135 140
Gly Arg Asn Asn Phe Asn Lys Phe Tyr Phe Gly Ser Gly Thr Lys Leu
145 150 155 160
Asn Val Lys Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu
165 170 175
Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe
180 185 190
Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile
195 200 205
Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn
210 215 220
Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala
225 230 235 240
Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu
245 250 255
Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr
260 265 270
Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu
275 280 285
Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser
290 295 300
Ser
305
<210> 829
<211> 305
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 40 - альфа, цистеин-модифицированный
<400> 829
Met His Thr Ser Thr Phe Gln Asn Arg Pro Gln Leu Phe Leu Leu Ile
1 5 10 15
Trp Lys Lys Leu Val Pro Gly Asn Pro Phe Arg Arg Ser Trp Met Lys
20 25 30
Arg Glu Arg Glu Met Leu Leu Ile Thr Ser Met Leu Val Leu Trp Met
35 40 45
Gln Leu Ser Gln Val Asn Gly Gln Gln Val Met Gln Ile Pro Gln Tyr
50 55 60
Gln His Val Gln Glu Gly Glu Asp Phe Thr Thr Tyr Cys Asn Ser Ser
65 70 75 80
Thr Thr Leu Ser Asn Ile Gln Trp Tyr Lys Gln Arg Pro Gly Gly His
85 90 95
Pro Val Phe Leu Ile Gln Leu Val Lys Ser Gly Glu Val Lys Lys Gln
100 105 110
Lys Arg Leu Thr Phe Gln Phe Gly Glu Ala Lys Lys Asn Ser Ser Leu
115 120 125
His Ile Thr Ala Thr Gln Thr Thr Asp Val Gly Thr Tyr Phe Cys Ala
130 135 140
Gly Arg Asn Asn Phe Asn Lys Phe Tyr Phe Gly Ser Gly Thr Lys Leu
145 150 155 160
Asn Val Lys Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu
165 170 175
Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe
180 185 190
Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile
195 200 205
Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn
210 215 220
Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala
225 230 235 240
Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu
245 250 255
Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr
260 265 270
Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu
275 280 285
Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser
290 295 300
Ser
305
<210> 830
<211> 110
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 40 альфа, вариабельная область
<400> 830
Gly Gln Gln Val Met Gln Ile Pro Gln Tyr Gln His Val Gln Glu Gly
1 5 10 15
Glu Asp Phe Thr Thr Tyr Cys Asn Ser Ser Thr Thr Leu Ser Asn Ile
20 25 30
Gln Trp Tyr Lys Gln Arg Pro Gly Gly His Pro Val Phe Leu Ile Gln
35 40 45
Leu Val Lys Ser Gly Glu Val Lys Lys Gln Lys Arg Leu Thr Phe Gln
50 55 60
Phe Gly Glu Ala Lys Lys Asn Ser Ser Leu His Ile Thr Ala Thr Gln
65 70 75 80
Thr Thr Asp Val Gly Thr Tyr Phe Cys Ala Gly Arg Asn Asn Phe Asn
85 90 95
Lys Phe Tyr Phe Gly Ser Gly Thr Lys Leu Asn Val Lys Pro
100 105 110
<210> 831
<211> 7
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 40/ TCR 42 альфа CDR2
<400> 831
Leu Val Lys Ser Gly Glu Val
1 5
<210> 832
<211> 10
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 40 альфа CDR3
<400> 832
Ala Gly Arg Asn Asn Phe Asn Lys Phe Tyr
1 5 10
<210> 833
<211> 137
<212> Белок
<213> Mus musculus
<220>
<223> Трансмембранно-модифицированная/цистеин-модифицированная мышиная
константная область альфа
<400> 833
Asn Ile Gln Asn Pro Glu Pro Ala Val Tyr Gln Leu Lys Asp Pro Arg
1 5 10 15
Ser Gln Asp Ser Thr Leu Cys Leu Phe Thr Asp Phe Asp Ser Gln Ile
20 25 30
Asn Val Pro Lys Thr Met Glu Ser Gly Thr Phe Ile Thr Asp Lys Cys
35 40 45
Val Leu Asp Met Lys Ala Met Asp Ser Lys Ser Asn Gly Ala Ile Ala
50 55 60
Trp Ser Asn Gln Thr Ser Phe Thr Cys Gln Asp Ile Phe Lys Glu Thr
65 70 75 80
Asn Ala Thr Tyr Pro Ser Ser Asp Val Pro Cys Asp Ala Thr Leu Thr
85 90 95
Glu Lys Ser Phe Glu Thr Asp Met Asn Leu Asn Phe Gln Asn Leu Leu
100 105 110
Val Ile Val Leu Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu
115 120 125
Leu Met Thr Leu Arg Leu Trp Ser Ser
130 135
<210> 834
<211> 54
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 40/ TCR 42/ TCR 45 альфа, Сигнальный пептид
<400> 834
Met His Thr Ser Thr Phe Gln Asn Arg Pro Gln Leu Phe Leu Leu Ile
1 5 10 15
Trp Lys Lys Leu Val Pro Gly Asn Pro Phe Arg Arg Ser Trp Met Lys
20 25 30
Arg Glu Arg Glu Met Leu Leu Ile Thr Ser Met Leu Val Leu Trp Met
35 40 45
Gln Leu Ser Gln Val Asn
50
<210> 835
<211> 292
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 40 - бета нативный
<400> 835
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Ile Thr Ala
85 90 95
Arg Ser Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu
115 120 125
Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys
130 135 140
Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val
145 150 155 160
Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro Leu
165 170 175
Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg
180 185 190
Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg
195 200 205
Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln
210 215 220
Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly
225 230 235 240
Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu
245 250 255
Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr
260 265 270
Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys
275 280 285
Asp Ser Arg Gly
290
<210> 836
<211> 292
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 40 - бета, цистеин-модифицированный
<400> 836
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Ile Thr Ala
85 90 95
Arg Ser Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu
115 120 125
Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys
130 135 140
Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val
145 150 155 160
Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro Leu
165 170 175
Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg
180 185 190
Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg
195 200 205
Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln
210 215 220
Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly
225 230 235 240
Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu
245 250 255
Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr
260 265 270
Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys
275 280 285
Asp Ser Arg Gly
290
<210> 837
<211> 311
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 40 - бета нативный
<400> 837
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ile Thr Ala Arg Ser Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 838
<211> 311
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 40 - бета, цистеин-модифицированный
<400> 838
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ile Thr Ala Arg Ser Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 839
<211> 113
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 40 бета, вариабельная область
<400> 839
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Ile Thr Ala
85 90 95
Arg Ser Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Thr
<210> 840
<211> 11
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 40 бета CDR3
<400> 840
Ala Ile Thr Ala Arg Ser Ser Tyr Glu Gln Tyr
1 5 10
<210> 841
<211> 253
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 41 - альфа нативный
<400> 841
Arg Lys Glu Val Glu Gln Asp Pro Gly Pro Phe Asn Val Pro Glu Gly
1 5 10 15
Ala Thr Val Ala Phe Asn Cys Thr Tyr Ser Asn Ser Ala Ser Gln Ser
20 25 30
Phe Phe Trp Tyr Arg Gln Asp Cys Arg Lys Glu Pro Lys Leu Leu Met
35 40 45
Ser Val Tyr Ser Ser Gly Asn Glu Asp Gly Arg Phe Thr Ala Gln Leu
50 55 60
Asn Arg Ala Ser Gln Tyr Ile Ser Leu Leu Ile Arg Asp Ser Lys Leu
65 70 75 80
Ser Asp Ser Ala Thr Tyr Leu Cys Val Val Thr Phe Ala Leu Thr Gly
85 90 95
Gly Phe Lys Thr Ile Phe Gly Ala Gly Thr Arg Leu Phe Val Lys Ala
100 105 110
Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys
115 120 125
Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr
130 135 140
Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr
145 150 155 160
Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala
165 170 175
Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser
180 185 190
Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp
195 200 205
Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe
210 215 220
Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala
225 230 235 240
Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 842
<211> 253
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 41 - альфа
цистеин-модифицированный
<400> 842
Arg Lys Glu Val Glu Gln Asp Pro Gly Pro Phe Asn Val Pro Glu Gly
1 5 10 15
Ala Thr Val Ala Phe Asn Cys Thr Tyr Ser Asn Ser Ala Ser Gln Ser
20 25 30
Phe Phe Trp Tyr Arg Gln Asp Cys Arg Lys Glu Pro Lys Leu Leu Met
35 40 45
Ser Val Tyr Ser Ser Gly Asn Glu Asp Gly Arg Phe Thr Ala Gln Leu
50 55 60
Asn Arg Ala Ser Gln Tyr Ile Ser Leu Leu Ile Arg Asp Ser Lys Leu
65 70 75 80
Ser Asp Ser Ala Thr Tyr Leu Cys Val Val Thr Phe Ala Leu Thr Gly
85 90 95
Gly Phe Lys Thr Ile Phe Gly Ala Gly Thr Arg Leu Phe Val Lys Ala
100 105 110
Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys
115 120 125
Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr
130 135 140
Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys
145 150 155 160
Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala
165 170 175
Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser
180 185 190
Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp
195 200 205
Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe
210 215 220
Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala
225 230 235 240
Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 843
<211> 275
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 41 - альфа нативный
<400> 843
Met Met Ile Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu
1 5 10 15
Ser Trp Val Trp Ser Gln Arg Lys Glu Val Glu Gln Asp Pro Gly Pro
20 25 30
Phe Asn Val Pro Glu Gly Ala Thr Val Ala Phe Asn Cys Thr Tyr Ser
35 40 45
Asn Ser Ala Ser Gln Ser Phe Phe Trp Tyr Arg Gln Asp Cys Arg Lys
50 55 60
Glu Pro Lys Leu Leu Met Ser Val Tyr Ser Ser Gly Asn Glu Asp Gly
65 70 75 80
Arg Phe Thr Ala Gln Leu Asn Arg Ala Ser Gln Tyr Ile Ser Leu Leu
85 90 95
Ile Arg Asp Ser Lys Leu Ser Asp Ser Ala Thr Tyr Leu Cys Val Val
100 105 110
Thr Phe Ala Leu Thr Gly Gly Phe Lys Thr Ile Phe Gly Ala Gly Thr
115 120 125
Arg Leu Phe Val Lys Ala Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr
130 135 140
Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr
145 150 155 160
Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val
165 170 175
Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys
180 185 190
Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala
195 200 205
Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser
210 215 220
Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr
225 230 235 240
Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile
245 250 255
Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu
260 265 270
Trp Ser Ser
275
<210> 844
<211> 275
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 41 - альфа, цистеин-модифицированный
<400> 844
Met Met Ile Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu
1 5 10 15
Ser Trp Val Trp Ser Gln Arg Lys Glu Val Glu Gln Asp Pro Gly Pro
20 25 30
Phe Asn Val Pro Glu Gly Ala Thr Val Ala Phe Asn Cys Thr Tyr Ser
35 40 45
Asn Ser Ala Ser Gln Ser Phe Phe Trp Tyr Arg Gln Asp Cys Arg Lys
50 55 60
Glu Pro Lys Leu Leu Met Ser Val Tyr Ser Ser Gly Asn Glu Asp Gly
65 70 75 80
Arg Phe Thr Ala Gln Leu Asn Arg Ala Ser Gln Tyr Ile Ser Leu Leu
85 90 95
Ile Arg Asp Ser Lys Leu Ser Asp Ser Ala Thr Tyr Leu Cys Val Val
100 105 110
Thr Phe Ala Leu Thr Gly Gly Phe Lys Thr Ile Phe Gly Ala Gly Thr
115 120 125
Arg Leu Phe Val Lys Ala Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr
130 135 140
Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr
145 150 155 160
Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val
165 170 175
Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys
180 185 190
Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala
195 200 205
Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser
210 215 220
Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr
225 230 235 240
Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile
245 250 255
Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu
260 265 270
Trp Ser Ser
275
<210> 845
<211> 112
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 41 альфа, вариабельная область
<400> 845
Arg Lys Glu Val Glu Gln Asp Pro Gly Pro Phe Asn Val Pro Glu Gly
1 5 10 15
Ala Thr Val Ala Phe Asn Cys Thr Tyr Ser Asn Ser Ala Ser Gln Ser
20 25 30
Phe Phe Trp Tyr Arg Gln Asp Cys Arg Lys Glu Pro Lys Leu Leu Met
35 40 45
Ser Val Tyr Ser Ser Gly Asn Glu Asp Gly Arg Phe Thr Ala Gln Leu
50 55 60
Asn Arg Ala Ser Gln Tyr Ile Ser Leu Leu Ile Arg Asp Ser Lys Leu
65 70 75 80
Ser Asp Ser Ala Thr Tyr Leu Cys Val Val Thr Phe Ala Leu Thr Gly
85 90 95
Gly Phe Lys Thr Ile Phe Gly Ala Gly Thr Arg Leu Phe Val Lys Ala
100 105 110
<210> 846
<211> 13
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 41 альфа CDR3
<400> 846
Val Val Thr Phe Ala Leu Thr Gly Gly Phe Lys Thr Ile
1 5 10
<210> 847
<211> 294
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 41 - бета нативный
<400> 847
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Ser Asn Pro
85 90 95
Arg Asp Arg Val Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu
100 105 110
Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val
115 120 125
Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu
130 135 140
Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp
145 150 155 160
Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln
165 170 175
Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser
180 185 190
Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His
195 200 205
Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp
210 215 220
Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala
225 230 235 240
Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly
245 250 255
Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr
260 265 270
Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys
275 280 285
Arg Lys Asp Ser Arg Gly
290
<210> 848
<211> 294
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 41 - бета, цистеин-модифицированный
<400> 848
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Ser Asn Pro
85 90 95
Arg Asp Arg Val Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu
100 105 110
Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val
115 120 125
Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu
130 135 140
Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp
145 150 155 160
Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln
165 170 175
Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser
180 185 190
Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His
195 200 205
Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp
210 215 220
Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala
225 230 235 240
Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly
245 250 255
Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr
260 265 270
Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys
275 280 285
Arg Lys Asp Ser Arg Gly
290
<210> 849
<211> 313
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 41 - бета нативный
<400> 849
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ser Asn Pro Arg Asp Arg Val Ser Tyr Glu Gln Tyr Phe Gly Pro Gly
115 120 125
Thr Arg Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu
130 135 140
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
145 150 155 160
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu
165 170 175
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr
180 185 190
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
195 200 205
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
210 215 220
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
225 230 235 240
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
245 250 255
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr
260 265 270
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
275 280 285
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
290 295 300
Met Val Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 850
<211> 313
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 41 - бета
цистеин-модифицированный
<400> 850
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ser Asn Pro Arg Asp Arg Val Ser Tyr Glu Gln Tyr Phe Gly Pro Gly
115 120 125
Thr Arg Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu
130 135 140
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
145 150 155 160
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu
165 170 175
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr
180 185 190
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
195 200 205
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
210 215 220
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
225 230 235 240
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
245 250 255
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr
260 265 270
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
275 280 285
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
290 295 300
Met Val Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 851
<211> 115
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 41 бета, вариабельная область
<400> 851
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Ser Asn Pro
85 90 95
Arg Asp Arg Val Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu
100 105 110
Thr Val Thr
115
<210> 852
<211> 13
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 41 бета CDR3
<400> 852
Ala Ser Asn Pro Arg Asp Arg Val Ser Tyr Glu Gln Tyr
1 5 10
<210> 853
<211> 251
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 42 - альфа нативный
<400> 853
Gly Gln Gln Val Met Gln Ile Pro Gln Tyr Gln His Val Gln Glu Gly
1 5 10 15
Glu Asp Phe Thr Thr Tyr Cys Asn Ser Ser Thr Thr Leu Ser Asn Ile
20 25 30
Gln Trp Tyr Lys Gln Arg Pro Gly Gly His Pro Val Phe Leu Ile Gln
35 40 45
Leu Val Lys Ser Gly Glu Val Lys Lys Gln Lys Arg Leu Thr Phe Gln
50 55 60
Phe Gly Glu Ala Lys Lys Asn Ser Ser Leu His Ile Thr Ala Thr Gln
65 70 75 80
Thr Thr Asp Val Gly Thr Tyr Phe Cys Ala Gly Pro Asp Asn Phe Asn
85 90 95
Lys Phe Tyr Phe Gly Ser Gly Thr Lys Leu Asn Val Lys Pro Asn Ile
100 105 110
Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser
115 120 125
Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val
130 135 140
Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr Val Leu
145 150 155 160
Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser
165 170 175
Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile
180 185 190
Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys
195 200 205
Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn
210 215 220
Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe
225 230 235 240
Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 854
<211> 251
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 42 - альфа, цистеин-модифицированный
<400> 854
Gly Gln Gln Val Met Gln Ile Pro Gln Tyr Gln His Val Gln Glu Gly
1 5 10 15
Glu Asp Phe Thr Thr Tyr Cys Asn Ser Ser Thr Thr Leu Ser Asn Ile
20 25 30
Gln Trp Tyr Lys Gln Arg Pro Gly Gly His Pro Val Phe Leu Ile Gln
35 40 45
Leu Val Lys Ser Gly Glu Val Lys Lys Gln Lys Arg Leu Thr Phe Gln
50 55 60
Phe Gly Glu Ala Lys Lys Asn Ser Ser Leu His Ile Thr Ala Thr Gln
65 70 75 80
Thr Thr Asp Val Gly Thr Tyr Phe Cys Ala Gly Pro Asp Asn Phe Asn
85 90 95
Lys Phe Tyr Phe Gly Ser Gly Thr Lys Leu Asn Val Lys Pro Asn Ile
100 105 110
Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser
115 120 125
Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val
130 135 140
Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys Val Leu
145 150 155 160
Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser
165 170 175
Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile
180 185 190
Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys
195 200 205
Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn
210 215 220
Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe
225 230 235 240
Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 855
<211> 305
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 42 - альфа нативный
<400> 855
Met His Thr Ser Thr Phe Gln Asn Arg Pro Gln Leu Phe Leu Leu Ile
1 5 10 15
Trp Lys Lys Leu Val Pro Gly Asn Pro Phe Arg Arg Ser Trp Met Lys
20 25 30
Arg Glu Arg Glu Met Leu Leu Ile Thr Ser Met Leu Val Leu Trp Met
35 40 45
Gln Leu Ser Gln Val Asn Gly Gln Gln Val Met Gln Ile Pro Gln Tyr
50 55 60
Gln His Val Gln Glu Gly Glu Asp Phe Thr Thr Tyr Cys Asn Ser Ser
65 70 75 80
Thr Thr Leu Ser Asn Ile Gln Trp Tyr Lys Gln Arg Pro Gly Gly His
85 90 95
Pro Val Phe Leu Ile Gln Leu Val Lys Ser Gly Glu Val Lys Lys Gln
100 105 110
Lys Arg Leu Thr Phe Gln Phe Gly Glu Ala Lys Lys Asn Ser Ser Leu
115 120 125
His Ile Thr Ala Thr Gln Thr Thr Asp Val Gly Thr Tyr Phe Cys Ala
130 135 140
Gly Pro Asp Asn Phe Asn Lys Phe Tyr Phe Gly Ser Gly Thr Lys Leu
145 150 155 160
Asn Val Lys Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu
165 170 175
Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe
180 185 190
Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile
195 200 205
Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn
210 215 220
Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala
225 230 235 240
Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu
245 250 255
Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr
260 265 270
Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu
275 280 285
Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser
290 295 300
Ser
305
<210> 856
<211> 305
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 42 - альфа
цистеин-модифицированный
<400> 856
Met His Thr Ser Thr Phe Gln Asn Arg Pro Gln Leu Phe Leu Leu Ile
1 5 10 15
Trp Lys Lys Leu Val Pro Gly Asn Pro Phe Arg Arg Ser Trp Met Lys
20 25 30
Arg Glu Arg Glu Met Leu Leu Ile Thr Ser Met Leu Val Leu Trp Met
35 40 45
Gln Leu Ser Gln Val Asn Gly Gln Gln Val Met Gln Ile Pro Gln Tyr
50 55 60
Gln His Val Gln Glu Gly Glu Asp Phe Thr Thr Tyr Cys Asn Ser Ser
65 70 75 80
Thr Thr Leu Ser Asn Ile Gln Trp Tyr Lys Gln Arg Pro Gly Gly His
85 90 95
Pro Val Phe Leu Ile Gln Leu Val Lys Ser Gly Glu Val Lys Lys Gln
100 105 110
Lys Arg Leu Thr Phe Gln Phe Gly Glu Ala Lys Lys Asn Ser Ser Leu
115 120 125
His Ile Thr Ala Thr Gln Thr Thr Asp Val Gly Thr Tyr Phe Cys Ala
130 135 140
Gly Pro Asp Asn Phe Asn Lys Phe Tyr Phe Gly Ser Gly Thr Lys Leu
145 150 155 160
Asn Val Lys Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu
165 170 175
Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe
180 185 190
Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile
195 200 205
Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn
210 215 220
Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala
225 230 235 240
Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu
245 250 255
Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr
260 265 270
Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu
275 280 285
Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser
290 295 300
Ser
305
<210> 857
<211> 110
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 42 альфа, вариабельная область
<400> 857
Gly Gln Gln Val Met Gln Ile Pro Gln Tyr Gln His Val Gln Glu Gly
1 5 10 15
Glu Asp Phe Thr Thr Tyr Cys Asn Ser Ser Thr Thr Leu Ser Asn Ile
20 25 30
Gln Trp Tyr Lys Gln Arg Pro Gly Gly His Pro Val Phe Leu Ile Gln
35 40 45
Leu Val Lys Ser Gly Glu Val Lys Lys Gln Lys Arg Leu Thr Phe Gln
50 55 60
Phe Gly Glu Ala Lys Lys Asn Ser Ser Leu His Ile Thr Ala Thr Gln
65 70 75 80
Thr Thr Asp Val Gly Thr Tyr Phe Cys Ala Gly Pro Asp Asn Phe Asn
85 90 95
Lys Phe Tyr Phe Gly Ser Gly Thr Lys Leu Asn Val Lys Pro
100 105 110
<210> 858
<211> 10
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 42 альфа CDR3
<400> 858
Ala Gly Pro Asp Asn Phe Asn Lys Phe Tyr
1 5 10
<210> 859
<211> 292
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 42 - бета нативный
<400> 859
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Lys Thr Ser
85 90 95
Arg Ser Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu
115 120 125
Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys
130 135 140
Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val
145 150 155 160
Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro Leu
165 170 175
Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg
180 185 190
Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg
195 200 205
Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln
210 215 220
Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly
225 230 235 240
Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu
245 250 255
Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr
260 265 270
Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys
275 280 285
Asp Ser Arg Gly
290
<210> 860
<211> 292
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 42 - бета, цистеин-модифицированный
<400> 860
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Lys Thr Ser
85 90 95
Arg Ser Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu
115 120 125
Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys
130 135 140
Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val
145 150 155 160
Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro Leu
165 170 175
Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg
180 185 190
Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg
195 200 205
Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln
210 215 220
Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly
225 230 235 240
Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu
245 250 255
Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr
260 265 270
Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys
275 280 285
Asp Ser Arg Gly
290
<210> 861
<211> 311
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 42 - бета нативный
<400> 861
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Lys Thr Ser Arg Ser Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 862
<211> 311
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 42 - бета, цистеин-модифицированный
<400> 862
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Lys Thr Ser Arg Ser Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 863
<211> 113
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 42 бета, вариабельная область
<400> 863
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Lys Thr Ser
85 90 95
Arg Ser Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Thr
<210> 864
<211> 11
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 42 бета CDR3
<400> 864
Ala Lys Thr Ser Arg Ser Ser Tyr Glu Gln Tyr
1 5 10
<210> 865
<211> 254
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 43 - альфа нативный
<400> 865
Gln Lys Glu Val Glu Gln Asn Ser Gly Pro Leu Ser Val Pro Glu Gly
1 5 10 15
Ala Ile Ala Ser Leu Asn Cys Thr Tyr Ser Asp Arg Gly Ser Gln Ser
20 25 30
Phe Phe Trp Tyr Arg Gln Tyr Ser Gly Lys Ser Pro Glu Leu Ile Met
35 40 45
Phe Ile Tyr Ser Asn Gly Asp Lys Glu Asp Gly Arg Phe Thr Ala Gln
50 55 60
Leu Asn Lys Ala Ser Gln Tyr Val Ser Leu Leu Ile Arg Asp Ser Gln
65 70 75 80
Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala Val Asn Val Pro Thr Ser
85 90 95
Gly Thr Tyr Lys Tyr Ile Phe Gly Thr Gly Thr Arg Leu Lys Val Leu
100 105 110
Ala Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser
115 120 125
Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln
130 135 140
Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys
145 150 155 160
Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val
165 170 175
Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn
180 185 190
Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys
195 200 205
Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn
210 215 220
Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val
225 230 235 240
Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 866
<211> 254
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 43 - альфа
цистеин-модифицированный
<400> 866
Gln Lys Glu Val Glu Gln Asn Ser Gly Pro Leu Ser Val Pro Glu Gly
1 5 10 15
Ala Ile Ala Ser Leu Asn Cys Thr Tyr Ser Asp Arg Gly Ser Gln Ser
20 25 30
Phe Phe Trp Tyr Arg Gln Tyr Ser Gly Lys Ser Pro Glu Leu Ile Met
35 40 45
Phe Ile Tyr Ser Asn Gly Asp Lys Glu Asp Gly Arg Phe Thr Ala Gln
50 55 60
Leu Asn Lys Ala Ser Gln Tyr Val Ser Leu Leu Ile Arg Asp Ser Gln
65 70 75 80
Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala Val Asn Val Pro Thr Ser
85 90 95
Gly Thr Tyr Lys Tyr Ile Phe Gly Thr Gly Thr Arg Leu Lys Val Leu
100 105 110
Ala Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser
115 120 125
Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln
130 135 140
Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys
145 150 155 160
Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val
165 170 175
Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn
180 185 190
Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys
195 200 205
Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn
210 215 220
Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val
225 230 235 240
Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 867
<211> 276
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 43 - альфа нативный
<400> 867
Met Met Lys Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu
1 5 10 15
Ser Trp Val Trp Ser Gln Gln Lys Glu Val Glu Gln Asn Ser Gly Pro
20 25 30
Leu Ser Val Pro Glu Gly Ala Ile Ala Ser Leu Asn Cys Thr Tyr Ser
35 40 45
Asp Arg Gly Ser Gln Ser Phe Phe Trp Tyr Arg Gln Tyr Ser Gly Lys
50 55 60
Ser Pro Glu Leu Ile Met Phe Ile Tyr Ser Asn Gly Asp Lys Glu Asp
65 70 75 80
Gly Arg Phe Thr Ala Gln Leu Asn Lys Ala Ser Gln Tyr Val Ser Leu
85 90 95
Leu Ile Arg Asp Ser Gln Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala
100 105 110
Val Asn Val Pro Thr Ser Gly Thr Tyr Lys Tyr Ile Phe Gly Thr Gly
115 120 125
Thr Arg Leu Lys Val Leu Ala Asn Ile Gln Asn Pro Asp Pro Ala Val
130 135 140
Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe
145 150 155 160
Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp
165 170 175
Val Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe
180 185 190
Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys
195 200 205
Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro
210 215 220
Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu
225 230 235 240
Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg
245 250 255
Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg
260 265 270
Leu Trp Ser Ser
275
<210> 868
<211> 276
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 43 - альфа, цистеин-модифицированный
<400> 868
Met Met Lys Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu
1 5 10 15
Ser Trp Val Trp Ser Gln Gln Lys Glu Val Glu Gln Asn Ser Gly Pro
20 25 30
Leu Ser Val Pro Glu Gly Ala Ile Ala Ser Leu Asn Cys Thr Tyr Ser
35 40 45
Asp Arg Gly Ser Gln Ser Phe Phe Trp Tyr Arg Gln Tyr Ser Gly Lys
50 55 60
Ser Pro Glu Leu Ile Met Phe Ile Tyr Ser Asn Gly Asp Lys Glu Asp
65 70 75 80
Gly Arg Phe Thr Ala Gln Leu Asn Lys Ala Ser Gln Tyr Val Ser Leu
85 90 95
Leu Ile Arg Asp Ser Gln Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala
100 105 110
Val Asn Val Pro Thr Ser Gly Thr Tyr Lys Tyr Ile Phe Gly Thr Gly
115 120 125
Thr Arg Leu Lys Val Leu Ala Asn Ile Gln Asn Pro Asp Pro Ala Val
130 135 140
Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe
145 150 155 160
Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp
165 170 175
Val Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe
180 185 190
Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys
195 200 205
Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro
210 215 220
Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu
225 230 235 240
Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg
245 250 255
Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg
260 265 270
Leu Trp Ser Ser
275
<210> 869
<211> 113
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 43 альфа, вариабельная область
<400> 869
Gln Lys Glu Val Glu Gln Asn Ser Gly Pro Leu Ser Val Pro Glu Gly
1 5 10 15
Ala Ile Ala Ser Leu Asn Cys Thr Tyr Ser Asp Arg Gly Ser Gln Ser
20 25 30
Phe Phe Trp Tyr Arg Gln Tyr Ser Gly Lys Ser Pro Glu Leu Ile Met
35 40 45
Phe Ile Tyr Ser Asn Gly Asp Lys Glu Asp Gly Arg Phe Thr Ala Gln
50 55 60
Leu Asn Lys Ala Ser Gln Tyr Val Ser Leu Leu Ile Arg Asp Ser Gln
65 70 75 80
Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala Val Asn Val Pro Thr Ser
85 90 95
Gly Thr Tyr Lys Tyr Ile Phe Gly Thr Gly Thr Arg Leu Lys Val Leu
100 105 110
Ala
<210> 870
<211> 13
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 43 альфа CDR3
<400> 870
Ala Val Asn Val Pro Thr Ser Gly Thr Tyr Lys Tyr Ile
1 5 10
<210> 871
<211> 292
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 43 - бета нативный
<400> 871
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Ser Thr Pro
85 90 95
Arg Asp Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu
115 120 125
Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys
130 135 140
Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val
145 150 155 160
Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro Leu
165 170 175
Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg
180 185 190
Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg
195 200 205
Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln
210 215 220
Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly
225 230 235 240
Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu
245 250 255
Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr
260 265 270
Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys
275 280 285
Asp Ser Arg Gly
290
<210> 872
<211> 292
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 43 - бета
цистеин-модифицированный
<400> 872
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Ser Thr Pro
85 90 95
Arg Asp Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu
115 120 125
Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys
130 135 140
Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val
145 150 155 160
Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro Leu
165 170 175
Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg
180 185 190
Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg
195 200 205
Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln
210 215 220
Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly
225 230 235 240
Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu
245 250 255
Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr
260 265 270
Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys
275 280 285
Asp Ser Arg Gly
290
<210> 873
<211> 311
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 43 - бета нативный
<400> 873
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ser Thr Pro Arg Asp Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 874
<211> 311
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 43 - бета
цистеин-модифицированный
<400> 874
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ser Thr Pro Arg Asp Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 875
<211> 113
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 43 бета, вариабельная область
<400> 875
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Ser Thr Pro
85 90 95
Arg Asp Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Thr
<210> 876
<211> 11
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 43 бета CDR3
<400> 876
Ala Ser Thr Pro Arg Asp Ser Tyr Glu Gln Tyr
1 5 10
<210> 877
<211> 251
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 44 - альфа нативный
<400> 877
Gly Gln Asn Ile Asp Gln Pro Thr Glu Met Thr Ala Thr Glu Gly Ala
1 5 10 15
Ile Val Gln Ile Asn Cys Thr Tyr Gln Thr Ser Gly Phe Asn Gly Leu
20 25 30
Phe Trp Tyr Gln Gln His Ala Gly Glu Ala Pro Thr Phe Leu Ser Tyr
35 40 45
Asn Val Leu Asp Gly Leu Glu Glu Lys Gly Arg Phe Ser Ser Phe Leu
50 55 60
Ser Arg Ser Lys Gly Tyr Ser Tyr Leu Leu Leu Lys Glu Leu Gln Met
65 70 75 80
Lys Asp Ser Ala Ser Tyr Leu Cys Ala Gln Tyr Ser Gly Gly Tyr Gln
85 90 95
Lys Val Thr Phe Gly Thr Gly Thr Lys Leu Gln Val Ile Pro Asn Ile
100 105 110
Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser
115 120 125
Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val
130 135 140
Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr Val Leu
145 150 155 160
Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser
165 170 175
Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile
180 185 190
Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys
195 200 205
Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn
210 215 220
Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe
225 230 235 240
Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 878
<211> 251
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 44 - альфа, цистеин-модифицированный
<400> 878
Gly Gln Asn Ile Asp Gln Pro Thr Glu Met Thr Ala Thr Glu Gly Ala
1 5 10 15
Ile Val Gln Ile Asn Cys Thr Tyr Gln Thr Ser Gly Phe Asn Gly Leu
20 25 30
Phe Trp Tyr Gln Gln His Ala Gly Glu Ala Pro Thr Phe Leu Ser Tyr
35 40 45
Asn Val Leu Asp Gly Leu Glu Glu Lys Gly Arg Phe Ser Ser Phe Leu
50 55 60
Ser Arg Ser Lys Gly Tyr Ser Tyr Leu Leu Leu Lys Glu Leu Gln Met
65 70 75 80
Lys Asp Ser Ala Ser Tyr Leu Cys Ala Gln Tyr Ser Gly Gly Tyr Gln
85 90 95
Lys Val Thr Phe Gly Thr Gly Thr Lys Leu Gln Val Ile Pro Asn Ile
100 105 110
Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser
115 120 125
Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val
130 135 140
Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys Val Leu
145 150 155 160
Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser
165 170 175
Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile
180 185 190
Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys
195 200 205
Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn
210 215 220
Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe
225 230 235 240
Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 879
<211> 285
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 44 - альфа нативный
<400> 879
Met Ala Gln Glu Leu Gly Met Gln Cys Gln Ala Arg Gly Ile Leu Gln
1 5 10 15
Gln Met Trp Gly Val Phe Leu Leu Tyr Val Ser Met Lys Met Gly Gly
20 25 30
Thr Thr Gly Gln Asn Ile Asp Gln Pro Thr Glu Met Thr Ala Thr Glu
35 40 45
Gly Ala Ile Val Gln Ile Asn Cys Thr Tyr Gln Thr Ser Gly Phe Asn
50 55 60
Gly Leu Phe Trp Tyr Gln Gln His Ala Gly Glu Ala Pro Thr Phe Leu
65 70 75 80
Ser Tyr Asn Val Leu Asp Gly Leu Glu Glu Lys Gly Arg Phe Ser Ser
85 90 95
Phe Leu Ser Arg Ser Lys Gly Tyr Ser Tyr Leu Leu Leu Lys Glu Leu
100 105 110
Gln Met Lys Asp Ser Ala Ser Tyr Leu Cys Ala Gln Tyr Ser Gly Gly
115 120 125
Tyr Gln Lys Val Thr Phe Gly Thr Gly Thr Lys Leu Gln Val Ile Pro
130 135 140
Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys
145 150 155 160
Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr
165 170 175
Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr
180 185 190
Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala
195 200 205
Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser
210 215 220
Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp
225 230 235 240
Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe
245 250 255
Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala
260 265 270
Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
275 280 285
<210> 880
<211> 285
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 44 - альфа
цистеин-модифицированный
<400> 880
Met Ala Gln Glu Leu Gly Met Gln Cys Gln Ala Arg Gly Ile Leu Gln
1 5 10 15
Gln Met Trp Gly Val Phe Leu Leu Tyr Val Ser Met Lys Met Gly Gly
20 25 30
Thr Thr Gly Gln Asn Ile Asp Gln Pro Thr Glu Met Thr Ala Thr Glu
35 40 45
Gly Ala Ile Val Gln Ile Asn Cys Thr Tyr Gln Thr Ser Gly Phe Asn
50 55 60
Gly Leu Phe Trp Tyr Gln Gln His Ala Gly Glu Ala Pro Thr Phe Leu
65 70 75 80
Ser Tyr Asn Val Leu Asp Gly Leu Glu Glu Lys Gly Arg Phe Ser Ser
85 90 95
Phe Leu Ser Arg Ser Lys Gly Tyr Ser Tyr Leu Leu Leu Lys Glu Leu
100 105 110
Gln Met Lys Asp Ser Ala Ser Tyr Leu Cys Ala Gln Tyr Ser Gly Gly
115 120 125
Tyr Gln Lys Val Thr Phe Gly Thr Gly Thr Lys Leu Gln Val Ile Pro
130 135 140
Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys
145 150 155 160
Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr
165 170 175
Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys
180 185 190
Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala
195 200 205
Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser
210 215 220
Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp
225 230 235 240
Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe
245 250 255
Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala
260 265 270
Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
275 280 285
<210> 881
<211> 110
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 44 альфа, вариабельная область
<400> 881
Gly Gln Asn Ile Asp Gln Pro Thr Glu Met Thr Ala Thr Glu Gly Ala
1 5 10 15
Ile Val Gln Ile Asn Cys Thr Tyr Gln Thr Ser Gly Phe Asn Gly Leu
20 25 30
Phe Trp Tyr Gln Gln His Ala Gly Glu Ala Pro Thr Phe Leu Ser Tyr
35 40 45
Asn Val Leu Asp Gly Leu Glu Glu Lys Gly Arg Phe Ser Ser Phe Leu
50 55 60
Ser Arg Ser Lys Gly Tyr Ser Tyr Leu Leu Leu Lys Glu Leu Gln Met
65 70 75 80
Lys Asp Ser Ala Ser Tyr Leu Cys Ala Gln Tyr Ser Gly Gly Tyr Gln
85 90 95
Lys Val Thr Phe Gly Thr Gly Thr Lys Leu Gln Val Ile Pro
100 105 110
<210> 882
<211> 11
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 44 альфа CDR3
<400> 882
Ala Gln Tyr Ser Gly Gly Tyr Gln Lys Val Thr
1 5 10
<210> 883
<211> 290
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 44 - бета нативный
<400> 883
Asp Ser Gly Val Thr Gln Thr Pro Lys His Leu Ile Thr Ala Thr Gly
1 5 10 15
Gln Arg Val Thr Leu Arg Cys Ser Pro Arg Ser Gly Asp Leu Ser Val
20 25 30
Tyr Trp Tyr Gln Gln Ser Leu Asp Gln Gly Leu Gln Phe Leu Ile Gln
35 40 45
Tyr Tyr Asn Gly Glu Glu Arg Ala Lys Gly Asn Ile Leu Glu Arg Phe
50 55 60
Ser Ala Gln Gln Phe Pro Asp Leu His Ser Glu Leu Asn Leu Ser Ser
65 70 75 80
Leu Glu Leu Gly Asp Ser Ala Leu Tyr Phe Cys Ala Ser Ser Gly Thr
85 90 95
Pro Asp Thr Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val Leu Glu
100 105 110
Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu Pro Ser
115 120 125
Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys Leu Ala
130 135 140
Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val Asn Gly
145 150 155 160
Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro Leu Lys Glu
165 170 175
Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg Leu Arg
180 185 190
Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg Cys Gln
195 200 205
Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln Asp Arg
210 215 220
Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly Arg Ala
225 230 235 240
Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu Ser Ala
245 250 255
Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr Ala Val
260 265 270
Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys Asp Ser
275 280 285
Arg Gly
290
<210> 884
<211> 290
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 44 - бета
цистеин-модифицированный
<400> 884
Asp Ser Gly Val Thr Gln Thr Pro Lys His Leu Ile Thr Ala Thr Gly
1 5 10 15
Gln Arg Val Thr Leu Arg Cys Ser Pro Arg Ser Gly Asp Leu Ser Val
20 25 30
Tyr Trp Tyr Gln Gln Ser Leu Asp Gln Gly Leu Gln Phe Leu Ile Gln
35 40 45
Tyr Tyr Asn Gly Glu Glu Arg Ala Lys Gly Asn Ile Leu Glu Arg Phe
50 55 60
Ser Ala Gln Gln Phe Pro Asp Leu His Ser Glu Leu Asn Leu Ser Ser
65 70 75 80
Leu Glu Leu Gly Asp Ser Ala Leu Tyr Phe Cys Ala Ser Ser Gly Thr
85 90 95
Pro Asp Thr Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val Leu Glu
100 105 110
Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu Pro Ser
115 120 125
Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys Leu Ala
130 135 140
Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val Asn Gly
145 150 155 160
Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro Leu Lys Glu
165 170 175
Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg Leu Arg
180 185 190
Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg Cys Gln
195 200 205
Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln Asp Arg
210 215 220
Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly Arg Ala
225 230 235 240
Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu Ser Ala
245 250 255
Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr Ala Val
260 265 270
Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys Asp Ser
275 280 285
Arg Gly
290
<210> 885
<211> 309
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 44 - бета нативный
<400> 885
Met Gly Phe Arg Leu Leu Cys Cys Val Ala Phe Cys Leu Leu Gly Ala
1 5 10 15
Gly Pro Val Asp Ser Gly Val Thr Gln Thr Pro Lys His Leu Ile Thr
20 25 30
Ala Thr Gly Gln Arg Val Thr Leu Arg Cys Ser Pro Arg Ser Gly Asp
35 40 45
Leu Ser Val Tyr Trp Tyr Gln Gln Ser Leu Asp Gln Gly Leu Gln Phe
50 55 60
Leu Ile Gln Tyr Tyr Asn Gly Glu Glu Arg Ala Lys Gly Asn Ile Leu
65 70 75 80
Glu Arg Phe Ser Ala Gln Gln Phe Pro Asp Leu His Ser Glu Leu Asn
85 90 95
Leu Ser Ser Leu Glu Leu Gly Asp Ser Ala Leu Tyr Phe Cys Ala Ser
100 105 110
Ser Gly Thr Pro Asp Thr Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr
115 120 125
Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe
130 135 140
Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val
145 150 155 160
Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp
165 170 175
Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro
180 185 190
Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser
195 200 205
Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe
210 215 220
Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr
225 230 235 240
Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp
245 250 255
Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val
260 265 270
Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu
275 280 285
Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg
290 295 300
Lys Asp Ser Arg Gly
305
<210> 886
<211> 309
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 44 - бета
цистеин-модифицированный
<400> 886
Met Gly Phe Arg Leu Leu Cys Cys Val Ala Phe Cys Leu Leu Gly Ala
1 5 10 15
Gly Pro Val Asp Ser Gly Val Thr Gln Thr Pro Lys His Leu Ile Thr
20 25 30
Ala Thr Gly Gln Arg Val Thr Leu Arg Cys Ser Pro Arg Ser Gly Asp
35 40 45
Leu Ser Val Tyr Trp Tyr Gln Gln Ser Leu Asp Gln Gly Leu Gln Phe
50 55 60
Leu Ile Gln Tyr Tyr Asn Gly Glu Glu Arg Ala Lys Gly Asn Ile Leu
65 70 75 80
Glu Arg Phe Ser Ala Gln Gln Phe Pro Asp Leu His Ser Glu Leu Asn
85 90 95
Leu Ser Ser Leu Glu Leu Gly Asp Ser Ala Leu Tyr Phe Cys Ala Ser
100 105 110
Ser Gly Thr Pro Asp Thr Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr
115 120 125
Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe
130 135 140
Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val
145 150 155 160
Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp
165 170 175
Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro
180 185 190
Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser
195 200 205
Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe
210 215 220
Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr
225 230 235 240
Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp
245 250 255
Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val
260 265 270
Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu
275 280 285
Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg
290 295 300
Lys Asp Ser Arg Gly
305
<210> 887
<211> 110
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 44 бета, вариабельная область
<400> 887
Asp Ser Gly Val Thr Gln Thr Pro Lys His Leu Ile Thr Ala Thr Gly
1 5 10 15
Gln Arg Val Thr Leu Arg Cys Ser Pro Arg Ser Gly Asp Leu Ser Val
20 25 30
Tyr Trp Tyr Gln Gln Ser Leu Asp Gln Gly Leu Gln Phe Leu Ile Gln
35 40 45
Tyr Tyr Asn Gly Glu Glu Arg Ala Lys Gly Asn Ile Leu Glu Arg Phe
50 55 60
Ser Ala Gln Gln Phe Pro Asp Leu His Ser Glu Leu Asn Leu Ser Ser
65 70 75 80
Leu Glu Leu Gly Asp Ser Ala Leu Tyr Phe Cys Ala Ser Ser Gly Thr
85 90 95
Pro Asp Thr Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
<210> 888
<211> 10
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 44 бета CDR3
<400> 888
Ala Ser Ser Gly Thr Pro Asp Thr Gln Tyr
1 5 10
<210> 889
<211> 180
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 44 нативный TCR бета, Константная область
<400> 889
Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu
1 5 10 15
Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys
20 25 30
Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val
35 40 45
Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro Leu
50 55 60
Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg
65 70 75 80
Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg
85 90 95
Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln
100 105 110
Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly
115 120 125
Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu
130 135 140
Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr
145 150 155 160
Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys
165 170 175
Asp Ser Arg Gly
180
<210> 890
<211> 180
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 44 TCR бета, Константная область
<400> 890
Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu
1 5 10 15
Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys
20 25 30
Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val
35 40 45
Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro Leu
50 55 60
Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg
65 70 75 80
Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg
85 90 95
Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln
100 105 110
Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly
115 120 125
Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu
130 135 140
Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr
145 150 155 160
Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys
165 170 175
Asp Ser Arg Gly
180
<210> 891
<211> 253
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 45 - альфа нативный
<400> 891
Gly Gln Gln Val Met Gln Ile Pro Gln Tyr Gln His Val Gln Glu Gly
1 5 10 15
Glu Asp Phe Thr Thr Tyr Cys Asn Ser Ser Thr Thr Leu Ser Asn Ile
20 25 30
Gln Trp Tyr Lys Gln Arg Pro Gly Gly His Pro Val Phe Leu Ile Gln
35 40 45
Leu Val Lys Ser Gly Glu Val Lys Lys Gln Lys Arg Leu Thr Phe Gln
50 55 60
Phe Gly Glu Ala Lys Lys Asn Ser Ser Leu His Ile Thr Ala Thr Gln
65 70 75 80
Thr Thr Asp Val Gly Thr Tyr Phe Cys Ala Gly Ser Ser Gly Ala Gly
85 90 95
Ser Tyr Gln Leu Thr Phe Gly Lys Gly Thr Lys Leu Ser Val Ile Pro
100 105 110
Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys
115 120 125
Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr
130 135 140
Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr
145 150 155 160
Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala
165 170 175
Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser
180 185 190
Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp
195 200 205
Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe
210 215 220
Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala
225 230 235 240
Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 892
<211> 253
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 45 - альфа, цистеин-модифицированный
<400> 892
Gly Gln Gln Val Met Gln Ile Pro Gln Tyr Gln His Val Gln Glu Gly
1 5 10 15
Glu Asp Phe Thr Thr Tyr Cys Asn Ser Ser Thr Thr Leu Ser Asn Ile
20 25 30
Gln Trp Tyr Lys Gln Arg Pro Gly Gly His Pro Val Phe Leu Ile Gln
35 40 45
Leu Val Lys Ser Gly Glu Val Lys Lys Gln Lys Arg Leu Thr Phe Gln
50 55 60
Phe Gly Glu Ala Lys Lys Asn Ser Ser Leu His Ile Thr Ala Thr Gln
65 70 75 80
Thr Thr Asp Val Gly Thr Tyr Phe Cys Ala Gly Ser Ser Gly Ala Gly
85 90 95
Ser Tyr Gln Leu Thr Phe Gly Lys Gly Thr Lys Leu Ser Val Ile Pro
100 105 110
Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys
115 120 125
Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr
130 135 140
Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys
145 150 155 160
Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala
165 170 175
Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser
180 185 190
Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp
195 200 205
Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe
210 215 220
Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala
225 230 235 240
Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 893
<211> 307
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 45 - альфа нативный
<400> 893
Met His Thr Ser Thr Phe Gln Asn Arg Pro Gln Leu Phe Leu Leu Ile
1 5 10 15
Trp Lys Lys Leu Val Pro Gly Asn Pro Phe Arg Arg Ser Trp Met Lys
20 25 30
Arg Glu Arg Glu Met Leu Leu Ile Thr Ser Met Leu Val Leu Trp Met
35 40 45
Gln Leu Ser Gln Val Asn Gly Gln Gln Val Met Gln Ile Pro Gln Tyr
50 55 60
Gln His Val Gln Glu Gly Glu Asp Phe Thr Thr Tyr Cys Asn Ser Ser
65 70 75 80
Thr Thr Leu Ser Asn Ile Gln Trp Tyr Lys Gln Arg Pro Gly Gly His
85 90 95
Pro Val Phe Leu Ile Gln Leu Val Lys Ser Gly Glu Val Lys Lys Gln
100 105 110
Lys Arg Leu Thr Phe Gln Phe Gly Glu Ala Lys Lys Asn Ser Ser Leu
115 120 125
His Ile Thr Ala Thr Gln Thr Thr Asp Val Gly Thr Tyr Phe Cys Ala
130 135 140
Gly Ser Ser Gly Ala Gly Ser Tyr Gln Leu Thr Phe Gly Lys Gly Thr
145 150 155 160
Lys Leu Ser Val Ile Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr
165 170 175
Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr
180 185 190
Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val
195 200 205
Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys
210 215 220
Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala
225 230 235 240
Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser
245 250 255
Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr
260 265 270
Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile
275 280 285
Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu
290 295 300
Trp Ser Ser
305
<210> 894
<211> 307
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 45 - альфа, цистеин-модифицированный
<400> 894
Met His Thr Ser Thr Phe Gln Asn Arg Pro Gln Leu Phe Leu Leu Ile
1 5 10 15
Trp Lys Lys Leu Val Pro Gly Asn Pro Phe Arg Arg Ser Trp Met Lys
20 25 30
Arg Glu Arg Glu Met Leu Leu Ile Thr Ser Met Leu Val Leu Trp Met
35 40 45
Gln Leu Ser Gln Val Asn Gly Gln Gln Val Met Gln Ile Pro Gln Tyr
50 55 60
Gln His Val Gln Glu Gly Glu Asp Phe Thr Thr Tyr Cys Asn Ser Ser
65 70 75 80
Thr Thr Leu Ser Asn Ile Gln Trp Tyr Lys Gln Arg Pro Gly Gly His
85 90 95
Pro Val Phe Leu Ile Gln Leu Val Lys Ser Gly Glu Val Lys Lys Gln
100 105 110
Lys Arg Leu Thr Phe Gln Phe Gly Glu Ala Lys Lys Asn Ser Ser Leu
115 120 125
His Ile Thr Ala Thr Gln Thr Thr Asp Val Gly Thr Tyr Phe Cys Ala
130 135 140
Gly Ser Ser Gly Ala Gly Ser Tyr Gln Leu Thr Phe Gly Lys Gly Thr
145 150 155 160
Lys Leu Ser Val Ile Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr
165 170 175
Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr
180 185 190
Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val
195 200 205
Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys
210 215 220
Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala
225 230 235 240
Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser
245 250 255
Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr
260 265 270
Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile
275 280 285
Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu
290 295 300
Trp Ser Ser
305
<210> 895
<211> 112
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 45 альфа, вариабельная область
<400> 895
Gly Gln Gln Val Met Gln Ile Pro Gln Tyr Gln His Val Gln Glu Gly
1 5 10 15
Glu Asp Phe Thr Thr Tyr Cys Asn Ser Ser Thr Thr Leu Ser Asn Ile
20 25 30
Gln Trp Tyr Lys Gln Arg Pro Gly Gly His Pro Val Phe Leu Ile Gln
35 40 45
Leu Val Lys Ser Gly Glu Val Lys Lys Gln Lys Arg Leu Thr Phe Gln
50 55 60
Phe Gly Glu Ala Lys Lys Asn Ser Ser Leu His Ile Thr Ala Thr Gln
65 70 75 80
Thr Thr Asp Val Gly Thr Tyr Phe Cys Ala Gly Ser Ser Gly Ala Gly
85 90 95
Ser Tyr Gln Leu Thr Phe Gly Lys Gly Thr Lys Leu Ser Val Ile Pro
100 105 110
<210> 896
<211> 12
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 45 альфа CDR3
<400> 896
Ala Gly Ser Ser Gly Ala Gly Ser Tyr Gln Leu Thr
1 5 10
<210> 897
<211> 294
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 45 - бета нативный
<400> 897
Gly Ala Gly Val Ser Gln Ser Pro Ser Asn Lys Val Thr Glu Lys Gly
1 5 10 15
Lys Asp Val Glu Leu Arg Cys Asp Pro Ile Ser Gly His Thr Ala Leu
20 25 30
Tyr Trp Tyr Arg Gln Arg Leu Gly Gln Gly Leu Glu Phe Leu Ile Tyr
35 40 45
Phe Gln Gly Asn Ser Ala Pro Asp Lys Ser Gly Leu Pro Ser Asp Arg
50 55 60
Phe Ser Ala Glu Arg Thr Gly Glu Ser Val Ser Thr Leu Thr Ile Gln
65 70 75 80
Arg Thr Gln Gln Glu Asp Ser Ala Val Tyr Leu Cys Ala Ser Ser Leu
85 90 95
Tyr Leu Gly Thr Thr Gly Glu Leu Phe Phe Gly Glu Gly Ser Arg Leu
100 105 110
Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val
115 120 125
Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu
130 135 140
Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp
145 150 155 160
Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln
165 170 175
Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser
180 185 190
Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His
195 200 205
Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp
210 215 220
Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala
225 230 235 240
Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly
245 250 255
Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr
260 265 270
Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys
275 280 285
Arg Lys Asp Ser Arg Gly
290
<210> 898
<211> 294
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 45 - бета, цистеин-модифицированный
<400> 898
Gly Ala Gly Val Ser Gln Ser Pro Ser Asn Lys Val Thr Glu Lys Gly
1 5 10 15
Lys Asp Val Glu Leu Arg Cys Asp Pro Ile Ser Gly His Thr Ala Leu
20 25 30
Tyr Trp Tyr Arg Gln Arg Leu Gly Gln Gly Leu Glu Phe Leu Ile Tyr
35 40 45
Phe Gln Gly Asn Ser Ala Pro Asp Lys Ser Gly Leu Pro Ser Asp Arg
50 55 60
Phe Ser Ala Glu Arg Thr Gly Glu Ser Val Ser Thr Leu Thr Ile Gln
65 70 75 80
Arg Thr Gln Gln Glu Asp Ser Ala Val Tyr Leu Cys Ala Ser Ser Leu
85 90 95
Tyr Leu Gly Thr Thr Gly Glu Leu Phe Phe Gly Glu Gly Ser Arg Leu
100 105 110
Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val
115 120 125
Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu
130 135 140
Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp
145 150 155 160
Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln
165 170 175
Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser
180 185 190
Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His
195 200 205
Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp
210 215 220
Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala
225 230 235 240
Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly
245 250 255
Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr
260 265 270
Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys
275 280 285
Arg Lys Asp Ser Arg Gly
290
<210> 899
<211> 313
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 45 - бета нативный
<400> 899
Met Gly Thr Arg Leu Leu Phe Trp Val Ala Phe Cys Leu Leu Gly Ala
1 5 10 15
Tyr His Thr Gly Ala Gly Val Ser Gln Ser Pro Ser Asn Lys Val Thr
20 25 30
Glu Lys Gly Lys Asp Val Glu Leu Arg Cys Asp Pro Ile Ser Gly His
35 40 45
Thr Ala Leu Tyr Trp Tyr Arg Gln Arg Leu Gly Gln Gly Leu Glu Phe
50 55 60
Leu Ile Tyr Phe Gln Gly Asn Ser Ala Pro Asp Lys Ser Gly Leu Pro
65 70 75 80
Ser Asp Arg Phe Ser Ala Glu Arg Thr Gly Glu Ser Val Ser Thr Leu
85 90 95
Thr Ile Gln Arg Thr Gln Gln Glu Asp Ser Ala Val Tyr Leu Cys Ala
100 105 110
Ser Ser Leu Tyr Leu Gly Thr Thr Gly Glu Leu Phe Phe Gly Glu Gly
115 120 125
Ser Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu
130 135 140
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
145 150 155 160
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu
165 170 175
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr
180 185 190
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
195 200 205
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
210 215 220
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
225 230 235 240
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
245 250 255
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr
260 265 270
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
275 280 285
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
290 295 300
Met Val Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 900
<211> 313
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 45 - бета
цистеин-модифицированный
<400> 900
Met Gly Thr Arg Leu Leu Phe Trp Val Ala Phe Cys Leu Leu Gly Ala
1 5 10 15
Tyr His Thr Gly Ala Gly Val Ser Gln Ser Pro Ser Asn Lys Val Thr
20 25 30
Glu Lys Gly Lys Asp Val Glu Leu Arg Cys Asp Pro Ile Ser Gly His
35 40 45
Thr Ala Leu Tyr Trp Tyr Arg Gln Arg Leu Gly Gln Gly Leu Glu Phe
50 55 60
Leu Ile Tyr Phe Gln Gly Asn Ser Ala Pro Asp Lys Ser Gly Leu Pro
65 70 75 80
Ser Asp Arg Phe Ser Ala Glu Arg Thr Gly Glu Ser Val Ser Thr Leu
85 90 95
Thr Ile Gln Arg Thr Gln Gln Glu Asp Ser Ala Val Tyr Leu Cys Ala
100 105 110
Ser Ser Leu Tyr Leu Gly Thr Thr Gly Glu Leu Phe Phe Gly Glu Gly
115 120 125
Ser Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu
130 135 140
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
145 150 155 160
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu
165 170 175
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr
180 185 190
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
195 200 205
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
210 215 220
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
225 230 235 240
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
245 250 255
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr
260 265 270
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
275 280 285
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
290 295 300
Met Val Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 901
<211> 115
<212> Белок
<213> Hom sapiens
<220>
<223> TCR 45 бета, вариабельная область
<400> 901
Gly Ala Gly Val Ser Gln Ser Pro Ser Asn Lys Val Thr Glu Lys Gly
1 5 10 15
Lys Asp Val Glu Leu Arg Cys Asp Pro Ile Ser Gly His Thr Ala Leu
20 25 30
Tyr Trp Tyr Arg Gln Arg Leu Gly Gln Gly Leu Glu Phe Leu Ile Tyr
35 40 45
Phe Gln Gly Asn Ser Ala Pro Asp Lys Ser Gly Leu Pro Ser Asp Arg
50 55 60
Phe Ser Ala Glu Arg Thr Gly Glu Ser Val Ser Thr Leu Thr Ile Gln
65 70 75 80
Arg Thr Gln Gln Glu Asp Ser Ala Val Tyr Leu Cys Ala Ser Ser Leu
85 90 95
Tyr Leu Gly Thr Thr Gly Glu Leu Phe Phe Gly Glu Gly Ser Arg Leu
100 105 110
Thr Val Leu
115
<210> 902
<211> 13
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 45 бета CDR3
<400> 902
Ala Ser Ser Leu Tyr Leu Gly Thr Thr Gly Glu Leu Phe
1 5 10
<210> 903
<211> 19
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 45 бета, Сигнальный пептид
<400> 903
Met Gly Thr Arg Leu Leu Phe Trp Val Ala Phe Cys Leu Leu Gly Ala
1 5 10 15
Tyr His Thr
<210> 904
<211> 255
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 46 - альфа нативный
<400> 904
Ala Gln Ser Val Thr Gln Leu Asp Ser Gln Val Pro Val Phe Glu Glu
1 5 10 15
Ala Pro Val Glu Leu Arg Cys Asn Tyr Ser Ser Ser Val Ser Val Tyr
20 25 30
Leu Phe Trp Tyr Val Gln Tyr Pro Asn Gln Gly Leu Gln Leu Leu Leu
35 40 45
Lys Tyr Leu Ser Gly Ser Thr Leu Val Lys Gly Ile Asn Gly Phe Glu
50 55 60
Ala Glu Phe Asn Lys Ser Gln Thr Ser Phe His Leu Arg Lys Pro Ser
65 70 75 80
Val His Ile Ser Asp Thr Ala Glu Tyr Phe Cys Ala Val Ser Pro Ser
85 90 95
Ser Gly Thr Tyr Lys Tyr Ile Phe Gly Thr Gly Thr Arg Leu Lys Val
100 105 110
Leu Ala Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp
115 120 125
Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser
130 135 140
Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp
145 150 155 160
Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala
165 170 175
Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn
180 185 190
Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser
195 200 205
Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu
210 215 220
Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys
225 230 235 240
Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250 255
<210> 905
<211> 255
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 46 - альфа, цистеин-модифицированный
<400> 905
Ala Gln Ser Val Thr Gln Leu Asp Ser Gln Val Pro Val Phe Glu Glu
1 5 10 15
Ala Pro Val Glu Leu Arg Cys Asn Tyr Ser Ser Ser Val Ser Val Tyr
20 25 30
Leu Phe Trp Tyr Val Gln Tyr Pro Asn Gln Gly Leu Gln Leu Leu Leu
35 40 45
Lys Tyr Leu Ser Gly Ser Thr Leu Val Lys Gly Ile Asn Gly Phe Glu
50 55 60
Ala Glu Phe Asn Lys Ser Gln Thr Ser Phe His Leu Arg Lys Pro Ser
65 70 75 80
Val His Ile Ser Asp Thr Ala Glu Tyr Phe Cys Ala Val Ser Pro Ser
85 90 95
Ser Gly Thr Tyr Lys Tyr Ile Phe Gly Thr Gly Thr Arg Leu Lys Val
100 105 110
Leu Ala Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp
115 120 125
Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser
130 135 140
Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp
145 150 155 160
Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala
165 170 175
Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn
180 185 190
Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser
195 200 205
Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu
210 215 220
Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys
225 230 235 240
Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250 255
<210> 906
<211> 274
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 46 - альфа нативный
<400> 906
Met Leu Leu Leu Leu Val Pro Ala Phe Gln Val Ile Phe Thr Leu Gly
1 5 10 15
Gly Thr Arg Ala Gln Ser Val Thr Gln Leu Asp Ser Gln Val Pro Val
20 25 30
Phe Glu Glu Ala Pro Val Glu Leu Arg Cys Asn Tyr Ser Ser Ser Val
35 40 45
Ser Val Tyr Leu Phe Trp Tyr Val Gln Tyr Pro Asn Gln Gly Leu Gln
50 55 60
Leu Leu Leu Lys Tyr Leu Ser Gly Ser Thr Leu Val Lys Gly Ile Asn
65 70 75 80
Gly Phe Glu Ala Glu Phe Asn Lys Ser Gln Thr Ser Phe His Leu Arg
85 90 95
Lys Pro Ser Val His Ile Ser Asp Thr Ala Glu Tyr Phe Cys Ala Val
100 105 110
Ser Pro Ser Ser Gly Thr Tyr Lys Tyr Ile Phe Gly Thr Gly Thr Arg
115 120 125
Leu Lys Val Leu Ala Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln
130 135 140
Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp
145 150 155 160
Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr
165 170 175
Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser
180 185 190
Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn
195 200 205
Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro
210 215 220
Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp
225 230 235 240
Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu
245 250 255
Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp
260 265 270
Ser Ser
<210> 907
<211> 274
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 46 - альфа, цистеин-модифицированный
<400> 907
Met Leu Leu Leu Leu Val Pro Ala Phe Gln Val Ile Phe Thr Leu Gly
1 5 10 15
Gly Thr Arg Ala Gln Ser Val Thr Gln Leu Asp Ser Gln Val Pro Val
20 25 30
Phe Glu Glu Ala Pro Val Glu Leu Arg Cys Asn Tyr Ser Ser Ser Val
35 40 45
Ser Val Tyr Leu Phe Trp Tyr Val Gln Tyr Pro Asn Gln Gly Leu Gln
50 55 60
Leu Leu Leu Lys Tyr Leu Ser Gly Ser Thr Leu Val Lys Gly Ile Asn
65 70 75 80
Gly Phe Glu Ala Glu Phe Asn Lys Ser Gln Thr Ser Phe His Leu Arg
85 90 95
Lys Pro Ser Val His Ile Ser Asp Thr Ala Glu Tyr Phe Cys Ala Val
100 105 110
Ser Pro Ser Ser Gly Thr Tyr Lys Tyr Ile Phe Gly Thr Gly Thr Arg
115 120 125
Leu Lys Val Leu Ala Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln
130 135 140
Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp
145 150 155 160
Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr
165 170 175
Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser
180 185 190
Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn
195 200 205
Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro
210 215 220
Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp
225 230 235 240
Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu
245 250 255
Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp
260 265 270
Ser Ser
<210> 908
<211> 114
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 46 альфа, вариабельная область
<400> 908
Ala Gln Ser Val Thr Gln Leu Asp Ser Gln Val Pro Val Phe Glu Glu
1 5 10 15
Ala Pro Val Glu Leu Arg Cys Asn Tyr Ser Ser Ser Val Ser Val Tyr
20 25 30
Leu Phe Trp Tyr Val Gln Tyr Pro Asn Gln Gly Leu Gln Leu Leu Leu
35 40 45
Lys Tyr Leu Ser Gly Ser Thr Leu Val Lys Gly Ile Asn Gly Phe Glu
50 55 60
Ala Glu Phe Asn Lys Ser Gln Thr Ser Phe His Leu Arg Lys Pro Ser
65 70 75 80
Val His Ile Ser Asp Thr Ala Glu Tyr Phe Cys Ala Val Ser Pro Ser
85 90 95
Ser Gly Thr Tyr Lys Tyr Ile Phe Gly Thr Gly Thr Arg Leu Lys Val
100 105 110
Leu Ala
<210> 909
<211> 6
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 46 альфа CDR1
<400> 909
Ser Ser Val Ser Val Tyr
1 5
<210> 910
<211> 8
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 46 альфа CDR2
<400> 910
Tyr Leu Ser Gly Ser Thr Leu Val
1 5
<210> 911
<211> 12
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 46 альфа CDR3
<400> 911
Ala Val Ser Pro Ser Ser Gly Thr Tyr Lys Tyr Ile
1 5 10
<210> 912
<211> 19
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 46 альфа, Сигнальный пептид
<400> 912
Met Leu Leu Leu Leu Val Pro Ala Phe Gln Val Ile Phe Thr Leu Gly
1 5 10 15
Gly Thr Arg
<210> 913
<211> 294
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 46 - бета нативный
<400> 913
Gly Ala Gly Val Ser Gln Thr Pro Ser Asn Lys Val Thr Glu Lys Gly
1 5 10 15
Lys Tyr Val Glu Leu Arg Cys Asp Pro Ile Ser Gly His Thr Ala Leu
20 25 30
Tyr Trp Tyr Arg Gln Ser Leu Gly Gln Gly Pro Glu Phe Leu Ile Tyr
35 40 45
Phe Gln Gly Thr Gly Ala Ala Asp Asp Ser Gly Leu Pro Asn Asp Arg
50 55 60
Phe Phe Ala Val Arg Pro Glu Gly Ser Val Ser Thr Leu Lys Ile Gln
65 70 75 80
Arg Thr Glu Arg Gly Asp Ser Ala Val Tyr Leu Cys Ala Ser Ser Leu
85 90 95
Tyr Leu Gly Gly Ser Glu Thr Gln Tyr Phe Gly Pro Gly Thr Arg Leu
100 105 110
Leu Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val
115 120 125
Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu
130 135 140
Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp
145 150 155 160
Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln
165 170 175
Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser
180 185 190
Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His
195 200 205
Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp
210 215 220
Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala
225 230 235 240
Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly
245 250 255
Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr
260 265 270
Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys
275 280 285
Arg Lys Asp Ser Arg Gly
290
<210> 914
<211> 294
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 46 - бета, цистеин-модифицированный
<400> 914
Gly Ala Gly Val Ser Gln Thr Pro Ser Asn Lys Val Thr Glu Lys Gly
1 5 10 15
Lys Tyr Val Glu Leu Arg Cys Asp Pro Ile Ser Gly His Thr Ala Leu
20 25 30
Tyr Trp Tyr Arg Gln Ser Leu Gly Gln Gly Pro Glu Phe Leu Ile Tyr
35 40 45
Phe Gln Gly Thr Gly Ala Ala Asp Asp Ser Gly Leu Pro Asn Asp Arg
50 55 60
Phe Phe Ala Val Arg Pro Glu Gly Ser Val Ser Thr Leu Lys Ile Gln
65 70 75 80
Arg Thr Glu Arg Gly Asp Ser Ala Val Tyr Leu Cys Ala Ser Ser Leu
85 90 95
Tyr Leu Gly Gly Ser Glu Thr Gln Tyr Phe Gly Pro Gly Thr Arg Leu
100 105 110
Leu Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val
115 120 125
Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu
130 135 140
Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp
145 150 155 160
Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln
165 170 175
Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser
180 185 190
Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His
195 200 205
Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp
210 215 220
Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala
225 230 235 240
Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly
245 250 255
Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr
260 265 270
Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys
275 280 285
Arg Lys Asp Ser Arg Gly
290
<210> 915
<211> 313
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 46 - бета нативный
<400> 915
Met Gly Thr Arg Leu Leu Cys Trp Ala Ala Leu Cys Leu Leu Gly Ala
1 5 10 15
Asp His Thr Gly Ala Gly Val Ser Gln Thr Pro Ser Asn Lys Val Thr
20 25 30
Glu Lys Gly Lys Tyr Val Glu Leu Arg Cys Asp Pro Ile Ser Gly His
35 40 45
Thr Ala Leu Tyr Trp Tyr Arg Gln Ser Leu Gly Gln Gly Pro Glu Phe
50 55 60
Leu Ile Tyr Phe Gln Gly Thr Gly Ala Ala Asp Asp Ser Gly Leu Pro
65 70 75 80
Asn Asp Arg Phe Phe Ala Val Arg Pro Glu Gly Ser Val Ser Thr Leu
85 90 95
Lys Ile Gln Arg Thr Glu Arg Gly Asp Ser Ala Val Tyr Leu Cys Ala
100 105 110
Ser Ser Leu Tyr Leu Gly Gly Ser Glu Thr Gln Tyr Phe Gly Pro Gly
115 120 125
Thr Arg Leu Leu Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu
130 135 140
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
145 150 155 160
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu
165 170 175
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr
180 185 190
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
195 200 205
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
210 215 220
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
225 230 235 240
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
245 250 255
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr
260 265 270
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
275 280 285
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
290 295 300
Met Val Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 916
<211> 313
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 46 - бета
цистеин-модифицированный
<400> 916
Met Gly Thr Arg Leu Leu Cys Trp Ala Ala Leu Cys Leu Leu Gly Ala
1 5 10 15
Asp His Thr Gly Ala Gly Val Ser Gln Thr Pro Ser Asn Lys Val Thr
20 25 30
Glu Lys Gly Lys Tyr Val Glu Leu Arg Cys Asp Pro Ile Ser Gly His
35 40 45
Thr Ala Leu Tyr Trp Tyr Arg Gln Ser Leu Gly Gln Gly Pro Glu Phe
50 55 60
Leu Ile Tyr Phe Gln Gly Thr Gly Ala Ala Asp Asp Ser Gly Leu Pro
65 70 75 80
Asn Asp Arg Phe Phe Ala Val Arg Pro Glu Gly Ser Val Ser Thr Leu
85 90 95
Lys Ile Gln Arg Thr Glu Arg Gly Asp Ser Ala Val Tyr Leu Cys Ala
100 105 110
Ser Ser Leu Tyr Leu Gly Gly Ser Glu Thr Gln Tyr Phe Gly Pro Gly
115 120 125
Thr Arg Leu Leu Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu
130 135 140
Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys
145 150 155 160
Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu
165 170 175
Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr
180 185 190
Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr
195 200 205
Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro
210 215 220
Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn
225 230 235 240
Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser
245 250 255
Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr
260 265 270
Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly
275 280 285
Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala
290 295 300
Met Val Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 917
<211> 115
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 46 бета, вариабельная область
<400> 917
Gly Ala Gly Val Ser Gln Thr Pro Ser Asn Lys Val Thr Glu Lys Gly
1 5 10 15
Lys Tyr Val Glu Leu Arg Cys Asp Pro Ile Ser Gly His Thr Ala Leu
20 25 30
Tyr Trp Tyr Arg Gln Ser Leu Gly Gln Gly Pro Glu Phe Leu Ile Tyr
35 40 45
Phe Gln Gly Thr Gly Ala Ala Asp Asp Ser Gly Leu Pro Asn Asp Arg
50 55 60
Phe Phe Ala Val Arg Pro Glu Gly Ser Val Ser Thr Leu Lys Ile Gln
65 70 75 80
Arg Thr Glu Arg Gly Asp Ser Ala Val Tyr Leu Cys Ala Ser Ser Leu
85 90 95
Tyr Leu Gly Gly Ser Glu Thr Gln Tyr Phe Gly Pro Gly Thr Arg Leu
100 105 110
Leu Val Leu
115
<210> 918
<211> 6
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 46 бета CDR2
<400> 918
Phe Gln Gly Thr Gly Ala
1 5
<210> 919
<211> 13
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 46 бета CDR3
<400> 919
Ala Ser Ser Leu Tyr Leu Gly Gly Ser Glu Thr Gln Tyr
1 5 10
<210> 920
<211> 19
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 46 бета, Сигнальный пептид
<400> 920
Met Gly Thr Arg Leu Leu Cys Trp Ala Ala Leu Cys Leu Leu Gly Ala
1 5 10 15
Asp His Thr
<210> 921
<211> 254
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 47 - альфа нативный
<400> 921
Gln Lys Glu Val Glu Gln Asn Ser Gly Pro Leu Ser Val Pro Glu Gly
1 5 10 15
Ala Ile Ala Ser Leu Asn Cys Thr Tyr Ser Asp Arg Gly Ser Gln Ser
20 25 30
Phe Phe Trp Tyr Arg Gln Tyr Ser Gly Lys Ser Pro Glu Leu Ile Met
35 40 45
Phe Ile Tyr Ser Asn Gly Asp Lys Glu Asp Gly Arg Phe Thr Ala Gln
50 55 60
Leu Asn Lys Ala Ser Gln Tyr Val Ser Leu Leu Ile Arg Asp Ser Gln
65 70 75 80
Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala Val Asn Leu Leu Ser Gly
85 90 95
Ser Ala Arg Gln Leu Thr Phe Gly Ser Gly Thr Gln Leu Thr Val Leu
100 105 110
Pro Asp Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser
115 120 125
Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln
130 135 140
Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys
145 150 155 160
Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val
165 170 175
Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn
180 185 190
Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys
195 200 205
Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn
210 215 220
Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val
225 230 235 240
Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 922
<211> 254
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 47 - альфа, цистеин-модифицированный
<400> 922
Gln Lys Glu Val Glu Gln Asn Ser Gly Pro Leu Ser Val Pro Glu Gly
1 5 10 15
Ala Ile Ala Ser Leu Asn Cys Thr Tyr Ser Asp Arg Gly Ser Gln Ser
20 25 30
Phe Phe Trp Tyr Arg Gln Tyr Ser Gly Lys Ser Pro Glu Leu Ile Met
35 40 45
Phe Ile Tyr Ser Asn Gly Asp Lys Glu Asp Gly Arg Phe Thr Ala Gln
50 55 60
Leu Asn Lys Ala Ser Gln Tyr Val Ser Leu Leu Ile Arg Asp Ser Gln
65 70 75 80
Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala Val Asn Leu Leu Ser Gly
85 90 95
Ser Ala Arg Gln Leu Thr Phe Gly Ser Gly Thr Gln Leu Thr Val Leu
100 105 110
Pro Asp Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser
115 120 125
Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln
130 135 140
Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys
145 150 155 160
Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val
165 170 175
Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn
180 185 190
Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys
195 200 205
Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn
210 215 220
Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val
225 230 235 240
Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 923
<211> 276
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 47 - альфа нативный
<400> 923
Met Met Lys Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu
1 5 10 15
Ser Trp Val Trp Ser Gln Gln Lys Glu Val Glu Gln Asn Ser Gly Pro
20 25 30
Leu Ser Val Pro Glu Gly Ala Ile Ala Ser Leu Asn Cys Thr Tyr Ser
35 40 45
Asp Arg Gly Ser Gln Ser Phe Phe Trp Tyr Arg Gln Tyr Ser Gly Lys
50 55 60
Ser Pro Glu Leu Ile Met Phe Ile Tyr Ser Asn Gly Asp Lys Glu Asp
65 70 75 80
Gly Arg Phe Thr Ala Gln Leu Asn Lys Ala Ser Gln Tyr Val Ser Leu
85 90 95
Leu Ile Arg Asp Ser Gln Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala
100 105 110
Val Asn Leu Leu Ser Gly Ser Ala Arg Gln Leu Thr Phe Gly Ser Gly
115 120 125
Thr Gln Leu Thr Val Leu Pro Asp Ile Gln Asn Pro Asp Pro Ala Val
130 135 140
Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe
145 150 155 160
Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp
165 170 175
Val Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe
180 185 190
Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys
195 200 205
Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro
210 215 220
Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu
225 230 235 240
Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg
245 250 255
Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg
260 265 270
Leu Trp Ser Ser
275
<210> 924
<211> 276
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 47 - альфа, цистеин-модифицированный
<400> 924
Met Met Lys Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu
1 5 10 15
Ser Trp Val Trp Ser Gln Gln Lys Glu Val Glu Gln Asn Ser Gly Pro
20 25 30
Leu Ser Val Pro Glu Gly Ala Ile Ala Ser Leu Asn Cys Thr Tyr Ser
35 40 45
Asp Arg Gly Ser Gln Ser Phe Phe Trp Tyr Arg Gln Tyr Ser Gly Lys
50 55 60
Ser Pro Glu Leu Ile Met Phe Ile Tyr Ser Asn Gly Asp Lys Glu Asp
65 70 75 80
Gly Arg Phe Thr Ala Gln Leu Asn Lys Ala Ser Gln Tyr Val Ser Leu
85 90 95
Leu Ile Arg Asp Ser Gln Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala
100 105 110
Val Asn Leu Leu Ser Gly Ser Ala Arg Gln Leu Thr Phe Gly Ser Gly
115 120 125
Thr Gln Leu Thr Val Leu Pro Asp Ile Gln Asn Pro Asp Pro Ala Val
130 135 140
Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe
145 150 155 160
Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp
165 170 175
Val Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe
180 185 190
Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys
195 200 205
Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro
210 215 220
Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu
225 230 235 240
Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg
245 250 255
Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg
260 265 270
Leu Trp Ser Ser
275
<210> 925
<211> 113
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 47 альфа, вариабельная область
<400> 925
Gln Lys Glu Val Glu Gln Asn Ser Gly Pro Leu Ser Val Pro Glu Gly
1 5 10 15
Ala Ile Ala Ser Leu Asn Cys Thr Tyr Ser Asp Arg Gly Ser Gln Ser
20 25 30
Phe Phe Trp Tyr Arg Gln Tyr Ser Gly Lys Ser Pro Glu Leu Ile Met
35 40 45
Phe Ile Tyr Ser Asn Gly Asp Lys Glu Asp Gly Arg Phe Thr Ala Gln
50 55 60
Leu Asn Lys Ala Ser Gln Tyr Val Ser Leu Leu Ile Arg Asp Ser Gln
65 70 75 80
Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala Val Asn Leu Leu Ser Gly
85 90 95
Ser Ala Arg Gln Leu Thr Phe Gly Ser Gly Thr Gln Leu Thr Val Leu
100 105 110
Pro
<210> 926
<211> 13
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 47 альфа CDR3
<400> 926
Ala Val Asn Leu Leu Ser Gly Ser Ala Arg Gln Leu Thr
1 5 10
<210> 927
<211> 292
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 47 - бета нативный
<400> 927
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Met Thr Gly
85 90 95
Arg Thr Thr Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu
115 120 125
Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys
130 135 140
Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val
145 150 155 160
Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro Leu
165 170 175
Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg
180 185 190
Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg
195 200 205
Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln
210 215 220
Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly
225 230 235 240
Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu
245 250 255
Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr
260 265 270
Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys
275 280 285
Asp Ser Arg Gly
290
<210> 928
<211> 292
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 47 - бета, цистеин-модифицированный
<400> 928
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Met Thr Gly
85 90 95
Arg Thr Thr Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu
115 120 125
Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys
130 135 140
Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val
145 150 155 160
Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro Leu
165 170 175
Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg
180 185 190
Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg
195 200 205
Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln
210 215 220
Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly
225 230 235 240
Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu
245 250 255
Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr
260 265 270
Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys
275 280 285
Asp Ser Arg Gly
290
<210> 929
<211> 311
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 47- бета нативный
<400> 929
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Met Thr Gly Arg Thr Thr Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 930
<211> 311
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 47 - бета
цистеин-модифицированный
<400> 930
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Met Thr Gly Arg Thr Thr Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 931
<211> 113
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 47 бета, вариабельная область
<400> 931
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Met Thr Gly
85 90 95
Arg Thr Thr Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Thr
<210> 932
<211> 11
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 47 бета CDR3
<400> 932
Ala Met Thr Gly Arg Thr Thr Tyr Glu Gln Tyr
1 5 10
<210> 933
<211> 252
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 48 - альфа нативный
<400> 933
Gln Lys Glu Val Glu Gln Asp Pro Gly Pro Leu Ser Val Pro Glu Gly
1 5 10 15
Ala Ile Val Ser Leu Asn Cys Thr Tyr Ser Asn Ser Ala Phe Gln Tyr
20 25 30
Phe Met Trp Tyr Arg Gln Tyr Ser Arg Lys Gly Pro Glu Leu Leu Met
35 40 45
Tyr Thr Tyr Ser Ser Gly Asn Lys Glu Asp Gly Arg Phe Thr Ala Gln
50 55 60
Val Asp Lys Ser Ser Lys Tyr Ile Ser Leu Phe Ile Arg Asp Ser Gln
65 70 75 80
Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala Met Arg Ile Gln Gly Ala
85 90 95
Gln Lys Leu Val Phe Gly Gln Gly Thr Arg Leu Thr Ile Asn Pro Asn
100 105 110
Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser
115 120 125
Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn
130 135 140
Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr Val
145 150 155 160
Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp
165 170 175
Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile
180 185 190
Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val
195 200 205
Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln
210 215 220
Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly
225 230 235 240
Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 934
<211> 252
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 48 - альфа
цистеин-модифицированный
<400> 934
Gln Lys Glu Val Glu Gln Asp Pro Gly Pro Leu Ser Val Pro Glu Gly
1 5 10 15
Ala Ile Val Ser Leu Asn Cys Thr Tyr Ser Asn Ser Ala Phe Gln Tyr
20 25 30
Phe Met Trp Tyr Arg Gln Tyr Ser Arg Lys Gly Pro Glu Leu Leu Met
35 40 45
Tyr Thr Tyr Ser Ser Gly Asn Lys Glu Asp Gly Arg Phe Thr Ala Gln
50 55 60
Val Asp Lys Ser Ser Lys Tyr Ile Ser Leu Phe Ile Arg Asp Ser Gln
65 70 75 80
Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala Met Arg Ile Gln Gly Ala
85 90 95
Gln Lys Leu Val Phe Gly Gln Gly Thr Arg Leu Thr Ile Asn Pro Asn
100 105 110
Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser
115 120 125
Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn
130 135 140
Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys Val
145 150 155 160
Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp
165 170 175
Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile
180 185 190
Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val
195 200 205
Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln
210 215 220
Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly
225 230 235 240
Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 935
<211> 274
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 48 - альфа нативный
<400> 935
Met Met Lys Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu
1 5 10 15
Ser Trp Val Trp Ser Gln Gln Lys Glu Val Glu Gln Asp Pro Gly Pro
20 25 30
Leu Ser Val Pro Glu Gly Ala Ile Val Ser Leu Asn Cys Thr Tyr Ser
35 40 45
Asn Ser Ala Phe Gln Tyr Phe Met Trp Tyr Arg Gln Tyr Ser Arg Lys
50 55 60
Gly Pro Glu Leu Leu Met Tyr Thr Tyr Ser Ser Gly Asn Lys Glu Asp
65 70 75 80
Gly Arg Phe Thr Ala Gln Val Asp Lys Ser Ser Lys Tyr Ile Ser Leu
85 90 95
Phe Ile Arg Asp Ser Gln Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala
100 105 110
Met Arg Ile Gln Gly Ala Gln Lys Leu Val Phe Gly Gln Gly Thr Arg
115 120 125
Leu Thr Ile Asn Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln
130 135 140
Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp
145 150 155 160
Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr
165 170 175
Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser
180 185 190
Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn
195 200 205
Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro
210 215 220
Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp
225 230 235 240
Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu
245 250 255
Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp
260 265 270
Ser Ser
<210> 936
<211> 274
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 48 - альфа
цистеин-модифицированный
<400> 936
Met Met Lys Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu
1 5 10 15
Ser Trp Val Trp Ser Gln Gln Lys Glu Val Glu Gln Asp Pro Gly Pro
20 25 30
Leu Ser Val Pro Glu Gly Ala Ile Val Ser Leu Asn Cys Thr Tyr Ser
35 40 45
Asn Ser Ala Phe Gln Tyr Phe Met Trp Tyr Arg Gln Tyr Ser Arg Lys
50 55 60
Gly Pro Glu Leu Leu Met Tyr Thr Tyr Ser Ser Gly Asn Lys Glu Asp
65 70 75 80
Gly Arg Phe Thr Ala Gln Val Asp Lys Ser Ser Lys Tyr Ile Ser Leu
85 90 95
Phe Ile Arg Asp Ser Gln Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala
100 105 110
Met Arg Ile Gln Gly Ala Gln Lys Leu Val Phe Gly Gln Gly Thr Arg
115 120 125
Leu Thr Ile Asn Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln
130 135 140
Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp
145 150 155 160
Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr
165 170 175
Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser
180 185 190
Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn
195 200 205
Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro
210 215 220
Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp
225 230 235 240
Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu
245 250 255
Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp
260 265 270
Ser Ser
<210> 937
<211> 111
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 48 альфа, вариабельная область
<400> 937
Gln Lys Glu Val Glu Gln Asp Pro Gly Pro Leu Ser Val Pro Glu Gly
1 5 10 15
Ala Ile Val Ser Leu Asn Cys Thr Tyr Ser Asn Ser Ala Phe Gln Tyr
20 25 30
Phe Met Trp Tyr Arg Gln Tyr Ser Arg Lys Gly Pro Glu Leu Leu Met
35 40 45
Tyr Thr Tyr Ser Ser Gly Asn Lys Glu Asp Gly Arg Phe Thr Ala Gln
50 55 60
Val Asp Lys Ser Ser Lys Tyr Ile Ser Leu Phe Ile Arg Asp Ser Gln
65 70 75 80
Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala Met Arg Ile Gln Gly Ala
85 90 95
Gln Lys Leu Val Phe Gly Gln Gly Thr Arg Leu Thr Ile Asn Pro
100 105 110
<210> 938
<211> 6
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 48 альфа CDR1
<400> 938
Asn Ser Ala Phe Gln Tyr
1 5
<210> 939
<211> 6
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 48 альфа CDR2
<400> 939
Thr Tyr Ser Ser Gly Asn
1 5
<210> 940
<211> 11
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 48 альфа CDR3
<400> 940
Ala Met Arg Ile Gln Gly Ala Gln Lys Leu Val
1 5 10
<210> 941
<211> 292
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 48 - бета нативный
<400> 941
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Ser Thr Gly
85 90 95
Arg Val Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu
115 120 125
Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys
130 135 140
Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val
145 150 155 160
Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro Leu
165 170 175
Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg
180 185 190
Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg
195 200 205
Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln
210 215 220
Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly
225 230 235 240
Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu
245 250 255
Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr
260 265 270
Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys
275 280 285
Asp Ser Arg Gly
290
<210> 942
<211> 292
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 48 - бета
цистеин-модифицированный
<400> 942
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Ser Thr Gly
85 90 95
Arg Val Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu
115 120 125
Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys
130 135 140
Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val
145 150 155 160
Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro Leu
165 170 175
Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg
180 185 190
Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg
195 200 205
Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln
210 215 220
Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly
225 230 235 240
Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu
245 250 255
Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr
260 265 270
Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys
275 280 285
Asp Ser Arg Gly
290
<210> 943
<211> 311
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 48- бета нативный
<400> 943
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ser Thr Gly Arg Val Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 944
<211> 311
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 48 - бета
цистеин-модифицированный
<400> 944
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ser Thr Gly Arg Val Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 945
<211> 113
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 48 бета, вариабельная область
<400> 945
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Ser Thr Gly
85 90 95
Arg Val Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Thr
<210> 946
<211> 11
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 48 бета CDR3
<400> 946
Ala Ser Thr Gly Arg Val Ser Tyr Glu Gln Tyr
1 5 10
<210> 947
<211> 254
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 49 - альфа нативный
<400> 947
Gln Lys Glu Val Glu Gln Asn Ser Gly Pro Leu Ser Val Pro Glu Gly
1 5 10 15
Ala Ile Ala Ser Leu Asn Cys Thr Tyr Ser Asp Arg Gly Ser Gln Ser
20 25 30
Phe Phe Trp Tyr Arg Gln Tyr Ser Gly Lys Ser Pro Glu Leu Ile Met
35 40 45
Phe Ile Tyr Ser Asn Gly Asp Lys Glu Asp Gly Arg Phe Thr Ala Gln
50 55 60
Leu Asn Lys Ala Ser Gln Tyr Val Ser Leu Leu Ile Arg Asp Ser Gln
65 70 75 80
Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala Val Asn Ile Gly Thr Ser
85 90 95
Gly Thr Tyr Lys Tyr Ile Phe Gly Thr Gly Thr Arg Leu Lys Val Leu
100 105 110
Ala Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser
115 120 125
Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln
130 135 140
Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys
145 150 155 160
Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val
165 170 175
Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn
180 185 190
Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys
195 200 205
Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn
210 215 220
Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val
225 230 235 240
Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 948
<211> 254
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 49 - альфа
цистеин-модифицированный
<400> 948
Gln Lys Glu Val Glu Gln Asn Ser Gly Pro Leu Ser Val Pro Glu Gly
1 5 10 15
Ala Ile Ala Ser Leu Asn Cys Thr Tyr Ser Asp Arg Gly Ser Gln Ser
20 25 30
Phe Phe Trp Tyr Arg Gln Tyr Ser Gly Lys Ser Pro Glu Leu Ile Met
35 40 45
Phe Ile Tyr Ser Asn Gly Asp Lys Glu Asp Gly Arg Phe Thr Ala Gln
50 55 60
Leu Asn Lys Ala Ser Gln Tyr Val Ser Leu Leu Ile Arg Asp Ser Gln
65 70 75 80
Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala Val Asn Ile Gly Thr Ser
85 90 95
Gly Thr Tyr Lys Tyr Ile Phe Gly Thr Gly Thr Arg Leu Lys Val Leu
100 105 110
Ala Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser
115 120 125
Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln
130 135 140
Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys
145 150 155 160
Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val
165 170 175
Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn
180 185 190
Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys
195 200 205
Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn
210 215 220
Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val
225 230 235 240
Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 949
<211> 276
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 49 - альфа нативный
<400> 949
Met Met Lys Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu
1 5 10 15
Ser Trp Val Trp Ser Gln Gln Lys Glu Val Glu Gln Asn Ser Gly Pro
20 25 30
Leu Ser Val Pro Glu Gly Ala Ile Ala Ser Leu Asn Cys Thr Tyr Ser
35 40 45
Asp Arg Gly Ser Gln Ser Phe Phe Trp Tyr Arg Gln Tyr Ser Gly Lys
50 55 60
Ser Pro Glu Leu Ile Met Phe Ile Tyr Ser Asn Gly Asp Lys Glu Asp
65 70 75 80
Gly Arg Phe Thr Ala Gln Leu Asn Lys Ala Ser Gln Tyr Val Ser Leu
85 90 95
Leu Ile Arg Asp Ser Gln Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala
100 105 110
Val Asn Ile Gly Thr Ser Gly Thr Tyr Lys Tyr Ile Phe Gly Thr Gly
115 120 125
Thr Arg Leu Lys Val Leu Ala Asn Ile Gln Asn Pro Asp Pro Ala Val
130 135 140
Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe
145 150 155 160
Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp
165 170 175
Val Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe
180 185 190
Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys
195 200 205
Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro
210 215 220
Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu
225 230 235 240
Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg
245 250 255
Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg
260 265 270
Leu Trp Ser Ser
275
<210> 950
<211> 276
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 49 - альфа
цистеин-модифицированный
<400> 950
Met Met Lys Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu
1 5 10 15
Ser Trp Val Trp Ser Gln Gln Lys Glu Val Glu Gln Asn Ser Gly Pro
20 25 30
Leu Ser Val Pro Glu Gly Ala Ile Ala Ser Leu Asn Cys Thr Tyr Ser
35 40 45
Asp Arg Gly Ser Gln Ser Phe Phe Trp Tyr Arg Gln Tyr Ser Gly Lys
50 55 60
Ser Pro Glu Leu Ile Met Phe Ile Tyr Ser Asn Gly Asp Lys Glu Asp
65 70 75 80
Gly Arg Phe Thr Ala Gln Leu Asn Lys Ala Ser Gln Tyr Val Ser Leu
85 90 95
Leu Ile Arg Asp Ser Gln Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala
100 105 110
Val Asn Ile Gly Thr Ser Gly Thr Tyr Lys Tyr Ile Phe Gly Thr Gly
115 120 125
Thr Arg Leu Lys Val Leu Ala Asn Ile Gln Asn Pro Asp Pro Ala Val
130 135 140
Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe
145 150 155 160
Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp
165 170 175
Val Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe
180 185 190
Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys
195 200 205
Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro
210 215 220
Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu
225 230 235 240
Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg
245 250 255
Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg
260 265 270
Leu Trp Ser Ser
275
<210> 951
<211> 113
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 49 альфа, вариабельная область
<400> 951
Gln Lys Glu Val Glu Gln Asn Ser Gly Pro Leu Ser Val Pro Glu Gly
1 5 10 15
Ala Ile Ala Ser Leu Asn Cys Thr Tyr Ser Asp Arg Gly Ser Gln Ser
20 25 30
Phe Phe Trp Tyr Arg Gln Tyr Ser Gly Lys Ser Pro Glu Leu Ile Met
35 40 45
Phe Ile Tyr Ser Asn Gly Asp Lys Glu Asp Gly Arg Phe Thr Ala Gln
50 55 60
Leu Asn Lys Ala Ser Gln Tyr Val Ser Leu Leu Ile Arg Asp Ser Gln
65 70 75 80
Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala Val Asn Ile Gly Thr Ser
85 90 95
Gly Thr Tyr Lys Tyr Ile Phe Gly Thr Gly Thr Arg Leu Lys Val Leu
100 105 110
Ala
<210> 952
<211> 13
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 49 альфа CDR3
<400> 952
Ala Val Asn Ile Gly Thr Ser Gly Thr Tyr Lys Tyr Ile
1 5 10
<210> 953
<211> 292
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 49 - бета нативный
<400> 953
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Ser Thr Pro
85 90 95
Arg Tyr Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu
115 120 125
Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys
130 135 140
Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val
145 150 155 160
Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro Leu
165 170 175
Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg
180 185 190
Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg
195 200 205
Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln
210 215 220
Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly
225 230 235 240
Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu
245 250 255
Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr
260 265 270
Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys
275 280 285
Asp Ser Arg Gly
290
<210> 954
<211> 292
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 49 - бета
цистеин-модифицированный
<400> 954
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Ser Thr Pro
85 90 95
Arg Tyr Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu
115 120 125
Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys
130 135 140
Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val
145 150 155 160
Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro Leu
165 170 175
Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg
180 185 190
Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg
195 200 205
Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln
210 215 220
Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly
225 230 235 240
Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu
245 250 255
Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr
260 265 270
Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys
275 280 285
Asp Ser Arg Gly
290
<210> 955
<211> 311
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 49- бета нативный
<400> 955
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ser Thr Pro Arg Tyr Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 956
<211> 311
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 49 - бета
цистеин-модифицированный
<400> 956
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ser Thr Pro Arg Tyr Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 957
<211> 113
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 49 бета, вариабельная область
<400> 957
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Ser Thr Pro
85 90 95
Arg Tyr Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Thr
<210> 958
<211> 11
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 49 бета CDR3
<400> 958
Ala Ser Thr Pro Arg Tyr Ser Tyr Glu Gln Tyr
1 5 10
<210> 959
<211> 252
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 50 - альфа нативный
<400> 959
Gly Glu Asp Val Glu Gln Ser Leu Phe Leu Ser Val Arg Glu Gly Asp
1 5 10 15
Ser Ser Val Ile Asn Cys Thr Tyr Thr Asp Ser Ser Ser Thr Tyr Leu
20 25 30
Tyr Trp Tyr Lys Gln Glu Pro Gly Ala Gly Leu Gln Leu Leu Thr Tyr
35 40 45
Ile Phe Ser Asn Met Asp Met Lys Gln Asp Gln Arg Leu Thr Val Leu
50 55 60
Leu Asn Lys Lys Asp Lys His Leu Ser Leu Arg Ile Ala Asp Thr Gln
65 70 75 80
Thr Gly Asp Ser Ala Ile Tyr Phe Cys Ala Glu Ser Pro Pro Gly Thr
85 90 95
Tyr Lys Tyr Ile Phe Gly Thr Gly Thr Arg Leu Lys Val Leu Ala Asn
100 105 110
Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser
115 120 125
Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn
130 135 140
Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr Val
145 150 155 160
Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp
165 170 175
Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile
180 185 190
Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val
195 200 205
Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln
210 215 220
Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly
225 230 235 240
Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 960
<211> 252
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 50 - альфа
цистеин-модифицированный
<400> 960
Gly Glu Asp Val Glu Gln Ser Leu Phe Leu Ser Val Arg Glu Gly Asp
1 5 10 15
Ser Ser Val Ile Asn Cys Thr Tyr Thr Asp Ser Ser Ser Thr Tyr Leu
20 25 30
Tyr Trp Tyr Lys Gln Glu Pro Gly Ala Gly Leu Gln Leu Leu Thr Tyr
35 40 45
Ile Phe Ser Asn Met Asp Met Lys Gln Asp Gln Arg Leu Thr Val Leu
50 55 60
Leu Asn Lys Lys Asp Lys His Leu Ser Leu Arg Ile Ala Asp Thr Gln
65 70 75 80
Thr Gly Asp Ser Ala Ile Tyr Phe Cys Ala Glu Ser Pro Pro Gly Thr
85 90 95
Tyr Lys Tyr Ile Phe Gly Thr Gly Thr Arg Leu Lys Val Leu Ala Asn
100 105 110
Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser
115 120 125
Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn
130 135 140
Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys Val
145 150 155 160
Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp
165 170 175
Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile
180 185 190
Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val
195 200 205
Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln
210 215 220
Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly
225 230 235 240
Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 961
<211> 273
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 50 - альфа нативный
<400> 961
Met Lys Thr Phe Ala Gly Phe Ser Phe Leu Phe Leu Trp Leu Gln Leu
1 5 10 15
Asp Cys Met Ser Arg Gly Glu Asp Val Glu Gln Ser Leu Phe Leu Ser
20 25 30
Val Arg Glu Gly Asp Ser Ser Val Ile Asn Cys Thr Tyr Thr Asp Ser
35 40 45
Ser Ser Thr Tyr Leu Tyr Trp Tyr Lys Gln Glu Pro Gly Ala Gly Leu
50 55 60
Gln Leu Leu Thr Tyr Ile Phe Ser Asn Met Asp Met Lys Gln Asp Gln
65 70 75 80
Arg Leu Thr Val Leu Leu Asn Lys Lys Asp Lys His Leu Ser Leu Arg
85 90 95
Ile Ala Asp Thr Gln Thr Gly Asp Ser Ala Ile Tyr Phe Cys Ala Glu
100 105 110
Ser Pro Pro Gly Thr Tyr Lys Tyr Ile Phe Gly Thr Gly Thr Arg Leu
115 120 125
Lys Val Leu Ala Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu
130 135 140
Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe
145 150 155 160
Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile
165 170 175
Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn
180 185 190
Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala
195 200 205
Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu
210 215 220
Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr
225 230 235 240
Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu
245 250 255
Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser
260 265 270
Ser
<210> 962
<211> 273
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 50 - альфа
цистеин-модифицированный
<400> 962
Met Lys Thr Phe Ala Gly Phe Ser Phe Leu Phe Leu Trp Leu Gln Leu
1 5 10 15
Asp Cys Met Ser Arg Gly Glu Asp Val Glu Gln Ser Leu Phe Leu Ser
20 25 30
Val Arg Glu Gly Asp Ser Ser Val Ile Asn Cys Thr Tyr Thr Asp Ser
35 40 45
Ser Ser Thr Tyr Leu Tyr Trp Tyr Lys Gln Glu Pro Gly Ala Gly Leu
50 55 60
Gln Leu Leu Thr Tyr Ile Phe Ser Asn Met Asp Met Lys Gln Asp Gln
65 70 75 80
Arg Leu Thr Val Leu Leu Asn Lys Lys Asp Lys His Leu Ser Leu Arg
85 90 95
Ile Ala Asp Thr Gln Thr Gly Asp Ser Ala Ile Tyr Phe Cys Ala Glu
100 105 110
Ser Pro Pro Gly Thr Tyr Lys Tyr Ile Phe Gly Thr Gly Thr Arg Leu
115 120 125
Lys Val Leu Ala Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu
130 135 140
Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe
145 150 155 160
Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile
165 170 175
Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn
180 185 190
Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala
195 200 205
Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu
210 215 220
Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr
225 230 235 240
Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu
245 250 255
Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser
260 265 270
Ser
<210> 963
<211> 111
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 50 альфа, вариабельная область
<400> 963
Gly Glu Asp Val Glu Gln Ser Leu Phe Leu Ser Val Arg Glu Gly Asp
1 5 10 15
Ser Ser Val Ile Asn Cys Thr Tyr Thr Asp Ser Ser Ser Thr Tyr Leu
20 25 30
Tyr Trp Tyr Lys Gln Glu Pro Gly Ala Gly Leu Gln Leu Leu Thr Tyr
35 40 45
Ile Phe Ser Asn Met Asp Met Lys Gln Asp Gln Arg Leu Thr Val Leu
50 55 60
Leu Asn Lys Lys Asp Lys His Leu Ser Leu Arg Ile Ala Asp Thr Gln
65 70 75 80
Thr Gly Asp Ser Ala Ile Tyr Phe Cys Ala Glu Ser Pro Pro Gly Thr
85 90 95
Tyr Lys Tyr Ile Phe Gly Thr Gly Thr Arg Leu Lys Val Leu Ala
100 105 110
<210> 964
<211> 11
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 50 альфа CDR3
<400> 964
Ala Glu Ser Pro Pro Gly Thr Tyr Lys Tyr Ile
1 5 10
<210> 965
<211> 290
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 50 - бета нативный
<400> 965
Asp Ser Gly Val Thr Gln Thr Pro Lys His Leu Ile Thr Ala Thr Gly
1 5 10 15
Gln Arg Val Thr Leu Arg Cys Ser Pro Arg Ser Gly Asp Leu Ser Val
20 25 30
Tyr Trp Tyr Gln Gln Ser Leu Asp Gln Gly Leu Gln Phe Leu Ile His
35 40 45
Tyr Tyr Asn Gly Glu Glu Arg Ala Lys Gly Asn Ile Leu Glu Arg Phe
50 55 60
Ser Ala Gln Gln Phe Pro Asp Leu His Ser Glu Leu Asn Leu Ser Ser
65 70 75 80
Leu Glu Leu Gly Asp Ser Ala Leu Tyr Phe Cys Ala Ser Ser Ala Thr
85 90 95
Arg Asp Ala Tyr Gly Tyr Thr Phe Gly Ser Gly Thr Arg Leu Thr Val
100 105 110
Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val Phe Glu
115 120 125
Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys
130 135 140
Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp Trp Val
145 150 155 160
Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro Leu
165 170 175
Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg
180 185 190
Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg
195 200 205
Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln
210 215 220
Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly
225 230 235 240
Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly Val Leu
245 250 255
Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr
260 265 270
Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys
275 280 285
Asp Phe
290
<210> 966
<211> 290
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 50 - бета
цистеин-модифицированный
<400> 966
Asp Ser Gly Val Thr Gln Thr Pro Lys His Leu Ile Thr Ala Thr Gly
1 5 10 15
Gln Arg Val Thr Leu Arg Cys Ser Pro Arg Ser Gly Asp Leu Ser Val
20 25 30
Tyr Trp Tyr Gln Gln Ser Leu Asp Gln Gly Leu Gln Phe Leu Ile His
35 40 45
Tyr Tyr Asn Gly Glu Glu Arg Ala Lys Gly Asn Ile Leu Glu Arg Phe
50 55 60
Ser Ala Gln Gln Phe Pro Asp Leu His Ser Glu Leu Asn Leu Ser Ser
65 70 75 80
Leu Glu Leu Gly Asp Ser Ala Leu Tyr Phe Cys Ala Ser Ser Ala Thr
85 90 95
Arg Asp Ala Tyr Gly Tyr Thr Phe Gly Ser Gly Thr Arg Leu Thr Val
100 105 110
Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val Phe Glu
115 120 125
Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys
130 135 140
Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp Trp Val
145 150 155 160
Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro Leu
165 170 175
Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg
180 185 190
Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg
195 200 205
Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln
210 215 220
Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly
225 230 235 240
Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly Val Leu
245 250 255
Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr
260 265 270
Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys
275 280 285
Asp Phe
290
<210> 967
<211> 309
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 50- бета нативный
<400> 967
Met Gly Phe Arg Leu Leu Cys Cys Val Ala Phe Cys Leu Leu Gly Ala
1 5 10 15
Gly Pro Val Asp Ser Gly Val Thr Gln Thr Pro Lys His Leu Ile Thr
20 25 30
Ala Thr Gly Gln Arg Val Thr Leu Arg Cys Ser Pro Arg Ser Gly Asp
35 40 45
Leu Ser Val Tyr Trp Tyr Gln Gln Ser Leu Asp Gln Gly Leu Gln Phe
50 55 60
Leu Ile His Tyr Tyr Asn Gly Glu Glu Arg Ala Lys Gly Asn Ile Leu
65 70 75 80
Glu Arg Phe Ser Ala Gln Gln Phe Pro Asp Leu His Ser Glu Leu Asn
85 90 95
Leu Ser Ser Leu Glu Leu Gly Asp Ser Ala Leu Tyr Phe Cys Ala Ser
100 105 110
Ser Ala Thr Arg Asp Ala Tyr Gly Tyr Thr Phe Gly Ser Gly Thr Arg
115 120 125
Leu Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Phe
305
<210> 968
<211> 309
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 50 - бета
цистеин-модифицированный
<400> 968
Met Gly Phe Arg Leu Leu Cys Cys Val Ala Phe Cys Leu Leu Gly Ala
1 5 10 15
Gly Pro Val Asp Ser Gly Val Thr Gln Thr Pro Lys His Leu Ile Thr
20 25 30
Ala Thr Gly Gln Arg Val Thr Leu Arg Cys Ser Pro Arg Ser Gly Asp
35 40 45
Leu Ser Val Tyr Trp Tyr Gln Gln Ser Leu Asp Gln Gly Leu Gln Phe
50 55 60
Leu Ile His Tyr Tyr Asn Gly Glu Glu Arg Ala Lys Gly Asn Ile Leu
65 70 75 80
Glu Arg Phe Ser Ala Gln Gln Phe Pro Asp Leu His Ser Glu Leu Asn
85 90 95
Leu Ser Ser Leu Glu Leu Gly Asp Ser Ala Leu Tyr Phe Cys Ala Ser
100 105 110
Ser Ala Thr Arg Asp Ala Tyr Gly Tyr Thr Phe Gly Ser Gly Thr Arg
115 120 125
Leu Thr Val Val Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Phe
305
<210> 969
<211> 113
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 50 бета, вариабельная область
<400> 969
Asp Ser Gly Val Thr Gln Thr Pro Lys His Leu Ile Thr Ala Thr Gly
1 5 10 15
Gln Arg Val Thr Leu Arg Cys Ser Pro Arg Ser Gly Asp Leu Ser Val
20 25 30
Tyr Trp Tyr Gln Gln Ser Leu Asp Gln Gly Leu Gln Phe Leu Ile His
35 40 45
Tyr Tyr Asn Gly Glu Glu Arg Ala Lys Gly Asn Ile Leu Glu Arg Phe
50 55 60
Ser Ala Gln Gln Phe Pro Asp Leu His Ser Glu Leu Asn Leu Ser Ser
65 70 75 80
Leu Glu Leu Gly Asp Ser Ala Leu Tyr Phe Cys Ala Ser Ser Ala Thr
85 90 95
Arg Asp Ala Tyr Gly Tyr Thr Phe Gly Ser Gly Thr Arg Leu Thr Val
100 105 110
Val
<210> 970
<211> 12
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 50 бета CDR3
<400> 970
Ala Ser Ser Ala Thr Arg Asp Ala Tyr Gly Tyr Thr
1 5 10
<210> 971
<211> 255
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 51 - альфа нативный
<400> 971
Gln Lys Glu Val Glu Gln Asn Ser Gly Pro Leu Ser Val Pro Glu Gly
1 5 10 15
Ala Ile Ala Ser Leu Asn Cys Thr Tyr Ser Asp Arg Gly Ser Gln Ser
20 25 30
Phe Phe Trp Tyr Arg Gln Tyr Ser Gly Lys Ser Pro Glu Leu Ile Met
35 40 45
Ser Ile Tyr Ser Asn Gly Asp Lys Glu Asp Gly Arg Phe Thr Ala Gln
50 55 60
Leu Asn Lys Ala Ser Gln Tyr Val Ser Leu Leu Ile Arg Asp Ser Gln
65 70 75 80
Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala Val Asn Met Arg Gly Gly
85 90 95
Gly Ser Asn Tyr Lys Leu Thr Phe Gly Lys Gly Thr Leu Leu Thr Val
100 105 110
Asn Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp
115 120 125
Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser
130 135 140
Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp
145 150 155 160
Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala
165 170 175
Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn
180 185 190
Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser
195 200 205
Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu
210 215 220
Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys
225 230 235 240
Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250 255
<210> 972
<211> 255
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 51 - альфа
цистеин-модифицированный
<400> 972
Gln Lys Glu Val Glu Gln Asn Ser Gly Pro Leu Ser Val Pro Glu Gly
1 5 10 15
Ala Ile Ala Ser Leu Asn Cys Thr Tyr Ser Asp Arg Gly Ser Gln Ser
20 25 30
Phe Phe Trp Tyr Arg Gln Tyr Ser Gly Lys Ser Pro Glu Leu Ile Met
35 40 45
Ser Ile Tyr Ser Asn Gly Asp Lys Glu Asp Gly Arg Phe Thr Ala Gln
50 55 60
Leu Asn Lys Ala Ser Gln Tyr Val Ser Leu Leu Ile Arg Asp Ser Gln
65 70 75 80
Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala Val Asn Met Arg Gly Gly
85 90 95
Gly Ser Asn Tyr Lys Leu Thr Phe Gly Lys Gly Thr Leu Leu Thr Val
100 105 110
Asn Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp
115 120 125
Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser
130 135 140
Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp
145 150 155 160
Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala
165 170 175
Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn
180 185 190
Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser
195 200 205
Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu
210 215 220
Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys
225 230 235 240
Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250 255
<210> 973
<211> 277
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 51 - альфа нативный
<400> 973
Met Met Lys Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu
1 5 10 15
Ser Trp Val Trp Ser Gln Gln Lys Glu Val Glu Gln Asn Ser Gly Pro
20 25 30
Leu Ser Val Pro Glu Gly Ala Ile Ala Ser Leu Asn Cys Thr Tyr Ser
35 40 45
Asp Arg Gly Ser Gln Ser Phe Phe Trp Tyr Arg Gln Tyr Ser Gly Lys
50 55 60
Ser Pro Glu Leu Ile Met Ser Ile Tyr Ser Asn Gly Asp Lys Glu Asp
65 70 75 80
Gly Arg Phe Thr Ala Gln Leu Asn Lys Ala Ser Gln Tyr Val Ser Leu
85 90 95
Leu Ile Arg Asp Ser Gln Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala
100 105 110
Val Asn Met Arg Gly Gly Gly Ser Asn Tyr Lys Leu Thr Phe Gly Lys
115 120 125
Gly Thr Leu Leu Thr Val Asn Pro Asn Ile Gln Asn Pro Asp Pro Ala
130 135 140
Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu
145 150 155 160
Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser
165 170 175
Asp Val Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp
180 185 190
Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala
195 200 205
Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe
210 215 220
Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe
225 230 235 240
Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe
245 250 255
Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu
260 265 270
Arg Leu Trp Ser Ser
275
<210> 974
<211> 277
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 51 - альфа
цистеин-модифицированный
<400> 974
Met Met Lys Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu
1 5 10 15
Ser Trp Val Trp Ser Gln Gln Lys Glu Val Glu Gln Asn Ser Gly Pro
20 25 30
Leu Ser Val Pro Glu Gly Ala Ile Ala Ser Leu Asn Cys Thr Tyr Ser
35 40 45
Asp Arg Gly Ser Gln Ser Phe Phe Trp Tyr Arg Gln Tyr Ser Gly Lys
50 55 60
Ser Pro Glu Leu Ile Met Ser Ile Tyr Ser Asn Gly Asp Lys Glu Asp
65 70 75 80
Gly Arg Phe Thr Ala Gln Leu Asn Lys Ala Ser Gln Tyr Val Ser Leu
85 90 95
Leu Ile Arg Asp Ser Gln Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala
100 105 110
Val Asn Met Arg Gly Gly Gly Ser Asn Tyr Lys Leu Thr Phe Gly Lys
115 120 125
Gly Thr Leu Leu Thr Val Asn Pro Asn Ile Gln Asn Pro Asp Pro Ala
130 135 140
Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu
145 150 155 160
Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser
165 170 175
Asp Val Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp
180 185 190
Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala
195 200 205
Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe
210 215 220
Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe
225 230 235 240
Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe
245 250 255
Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu
260 265 270
Arg Leu Trp Ser Ser
275
<210> 975
<211> 114
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 51 альфа, вариабельная область
<400> 975
Gln Lys Glu Val Glu Gln Asn Ser Gly Pro Leu Ser Val Pro Glu Gly
1 5 10 15
Ala Ile Ala Ser Leu Asn Cys Thr Tyr Ser Asp Arg Gly Ser Gln Ser
20 25 30
Phe Phe Trp Tyr Arg Gln Tyr Ser Gly Lys Ser Pro Glu Leu Ile Met
35 40 45
Ser Ile Tyr Ser Asn Gly Asp Lys Glu Asp Gly Arg Phe Thr Ala Gln
50 55 60
Leu Asn Lys Ala Ser Gln Tyr Val Ser Leu Leu Ile Arg Asp Ser Gln
65 70 75 80
Pro Ser Asp Ser Ala Thr Tyr Leu Cys Ala Val Asn Met Arg Gly Gly
85 90 95
Gly Ser Asn Tyr Lys Leu Thr Phe Gly Lys Gly Thr Leu Leu Thr Val
100 105 110
Asn Pro
<210> 976
<211> 14
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 51 альфа CDR3
<400> 976
Ala Val Asn Met Arg Gly Gly Gly Ser Asn Tyr Lys Leu Thr
1 5 10
<210> 977
<211> 292
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 51 - бета нативный
<400> 977
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Ile Ala Ser
85 90 95
Arg Val Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu
115 120 125
Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys
130 135 140
Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val
145 150 155 160
Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro Leu
165 170 175
Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg
180 185 190
Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg
195 200 205
Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln
210 215 220
Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly
225 230 235 240
Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu
245 250 255
Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr
260 265 270
Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys
275 280 285
Asp Ser Arg Gly
290
<210> 978
<211> 292
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 51 - бета
цистеин-модифицированный
<400> 978
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Ile Ala Ser
85 90 95
Arg Val Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu
115 120 125
Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys
130 135 140
Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val
145 150 155 160
Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro Leu
165 170 175
Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg
180 185 190
Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg
195 200 205
Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln
210 215 220
Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly
225 230 235 240
Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu
245 250 255
Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr
260 265 270
Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys
275 280 285
Asp Ser Arg Gly
290
<210> 979
<211> 311
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 51- бета нативный
<400> 979
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ile Ala Ser Arg Val Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 980
<211> 311
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 51 - бета
цистеин-модифицированный
<400> 980
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Ile Ala Ser Arg Val Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 981
<211> 113
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 51 бета, вариабельная область
<400> 981
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Ile Ala Ser
85 90 95
Arg Val Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Thr
<210> 982
<211> 11
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 51 бета CDR3
<400> 982
Ala Ile Ala Ser Arg Val Ser Tyr Glu Gln Tyr
1 5 10
<210> 983
<211> 250
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 52 - альфа нативный
<400> 983
Thr Gln Leu Leu Glu Gln Ser Pro Gln Phe Leu Ser Ile Gln Glu Gly
1 5 10 15
Glu Asn Leu Thr Val Tyr Cys Asn Ser Ser Ser Val Phe Ser Ser Leu
20 25 30
Gln Trp Tyr Arg Gln Glu Pro Gly Glu Gly Pro Val Leu Leu Val Thr
35 40 45
Val Val Thr Gly Gly Glu Val Lys Lys Leu Lys Arg Leu Thr Phe Gln
50 55 60
Phe Gly Asp Ala Arg Lys Asp Ser Ser Leu His Ile Thr Ala Ala Gln
65 70 75 80
Pro Gly Asp Thr Gly Leu Tyr Leu Cys Ala Gly Ala Arg Asp Tyr Lys
85 90 95
Leu Ser Phe Gly Ala Gly Thr Thr Val Thr Val Arg Ala Asn Ile Gln
100 105 110
Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp
115 120 125
Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser
130 135 140
Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr Val Leu Asp
145 150 155 160
Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn
165 170 175
Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro
180 185 190
Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu
195 200 205
Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu
210 215 220
Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn
225 230 235 240
Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 984
<211> 250
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 52 - альфа
цистеин-модифицированный
<400> 984
Thr Gln Leu Leu Glu Gln Ser Pro Gln Phe Leu Ser Ile Gln Glu Gly
1 5 10 15
Glu Asn Leu Thr Val Tyr Cys Asn Ser Ser Ser Val Phe Ser Ser Leu
20 25 30
Gln Trp Tyr Arg Gln Glu Pro Gly Glu Gly Pro Val Leu Leu Val Thr
35 40 45
Val Val Thr Gly Gly Glu Val Lys Lys Leu Lys Arg Leu Thr Phe Gln
50 55 60
Phe Gly Asp Ala Arg Lys Asp Ser Ser Leu His Ile Thr Ala Ala Gln
65 70 75 80
Pro Gly Asp Thr Gly Leu Tyr Leu Cys Ala Gly Ala Arg Asp Tyr Lys
85 90 95
Leu Ser Phe Gly Ala Gly Thr Thr Val Thr Val Arg Ala Asn Ile Gln
100 105 110
Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp
115 120 125
Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser
130 135 140
Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys Val Leu Asp
145 150 155 160
Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn
165 170 175
Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro
180 185 190
Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu
195 200 205
Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu
210 215 220
Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn
225 230 235 240
Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250
<210> 985
<211> 268
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 52 - альфа нативный
<400> 985
Met Val Leu Lys Phe Ser Val Ser Ile Leu Trp Ile Gln Leu Ala Trp
1 5 10 15
Val Ser Thr Gln Leu Leu Glu Gln Ser Pro Gln Phe Leu Ser Ile Gln
20 25 30
Glu Gly Glu Asn Leu Thr Val Tyr Cys Asn Ser Ser Ser Val Phe Ser
35 40 45
Ser Leu Gln Trp Tyr Arg Gln Glu Pro Gly Glu Gly Pro Val Leu Leu
50 55 60
Val Thr Val Val Thr Gly Gly Glu Val Lys Lys Leu Lys Arg Leu Thr
65 70 75 80
Phe Gln Phe Gly Asp Ala Arg Lys Asp Ser Ser Leu His Ile Thr Ala
85 90 95
Ala Gln Pro Gly Asp Thr Gly Leu Tyr Leu Cys Ala Gly Ala Arg Asp
100 105 110
Tyr Lys Leu Ser Phe Gly Ala Gly Thr Thr Val Thr Val Arg Ala Asn
115 120 125
Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser
130 135 140
Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn
145 150 155 160
Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr Val
165 170 175
Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp
180 185 190
Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile
195 200 205
Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val
210 215 220
Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln
225 230 235 240
Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly
245 250 255
Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
260 265
<210> 986
<211> 268
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 52 - альфа
цистеин-модифицированный
<400> 986
Met Val Leu Lys Phe Ser Val Ser Ile Leu Trp Ile Gln Leu Ala Trp
1 5 10 15
Val Ser Thr Gln Leu Leu Glu Gln Ser Pro Gln Phe Leu Ser Ile Gln
20 25 30
Glu Gly Glu Asn Leu Thr Val Tyr Cys Asn Ser Ser Ser Val Phe Ser
35 40 45
Ser Leu Gln Trp Tyr Arg Gln Glu Pro Gly Glu Gly Pro Val Leu Leu
50 55 60
Val Thr Val Val Thr Gly Gly Glu Val Lys Lys Leu Lys Arg Leu Thr
65 70 75 80
Phe Gln Phe Gly Asp Ala Arg Lys Asp Ser Ser Leu His Ile Thr Ala
85 90 95
Ala Gln Pro Gly Asp Thr Gly Leu Tyr Leu Cys Ala Gly Ala Arg Asp
100 105 110
Tyr Lys Leu Ser Phe Gly Ala Gly Thr Thr Val Thr Val Arg Ala Asn
115 120 125
Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser
130 135 140
Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn
145 150 155 160
Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Cys Val
165 170 175
Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala Trp
180 185 190
Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile
195 200 205
Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp Val
210 215 220
Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln
225 230 235 240
Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala Gly
245 250 255
Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
260 265
<210> 987
<211> 109
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 52 альфа, вариабельная область
<400> 987
Thr Gln Leu Leu Glu Gln Ser Pro Gln Phe Leu Ser Ile Gln Glu Gly
1 5 10 15
Glu Asn Leu Thr Val Tyr Cys Asn Ser Ser Ser Val Phe Ser Ser Leu
20 25 30
Gln Trp Tyr Arg Gln Glu Pro Gly Glu Gly Pro Val Leu Leu Val Thr
35 40 45
Val Val Thr Gly Gly Glu Val Lys Lys Leu Lys Arg Leu Thr Phe Gln
50 55 60
Phe Gly Asp Ala Arg Lys Asp Ser Ser Leu His Ile Thr Ala Ala Gln
65 70 75 80
Pro Gly Asp Thr Gly Leu Tyr Leu Cys Ala Gly Ala Arg Asp Tyr Lys
85 90 95
Leu Ser Phe Gly Ala Gly Thr Thr Val Thr Val Arg Ala
100 105
<210> 988
<211> 9
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 52 альфа CDR3
<400> 988
Ala Gly Ala Arg Asp Tyr Lys Leu Ser
1 5
<210> 989
<211> 292
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 52 - бета нативный
<400> 989
Asp Ser Gly Val Thr Gln Thr Pro Lys His Leu Ile Thr Ala Thr Gly
1 5 10 15
Gln Arg Val Thr Leu Arg Cys Ser Pro Arg Ser Gly Asp Leu Ser Val
20 25 30
Tyr Trp Tyr Gln Gln Ser Leu Asp Gln Gly Leu Gln Phe Leu Ile Gln
35 40 45
Tyr Tyr Asn Gly Glu Glu Arg Ala Lys Gly Asn Ile Leu Glu Arg Phe
50 55 60
Ser Ala Gln Gln Phe Pro Asp Leu His Ser Glu Leu Asn Leu Ser Ser
65 70 75 80
Leu Glu Leu Gly Asp Ser Ala Leu Tyr Phe Cys Ala Ser Ser Val Gly
85 90 95
Asp Leu Asn Asn Glu Gln Phe Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu
115 120 125
Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys
130 135 140
Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val
145 150 155 160
Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro Leu
165 170 175
Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg
180 185 190
Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg
195 200 205
Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln
210 215 220
Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly
225 230 235 240
Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu
245 250 255
Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr
260 265 270
Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys
275 280 285
Asp Ser Arg Gly
290
<210> 990
<211> 292
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 52 - бета, цистеин-модифицированный
<400> 990
Asp Ser Gly Val Thr Gln Thr Pro Lys His Leu Ile Thr Ala Thr Gly
1 5 10 15
Gln Arg Val Thr Leu Arg Cys Ser Pro Arg Ser Gly Asp Leu Ser Val
20 25 30
Tyr Trp Tyr Gln Gln Ser Leu Asp Gln Gly Leu Gln Phe Leu Ile Gln
35 40 45
Tyr Tyr Asn Gly Glu Glu Arg Ala Lys Gly Asn Ile Leu Glu Arg Phe
50 55 60
Ser Ala Gln Gln Phe Pro Asp Leu His Ser Glu Leu Asn Leu Ser Ser
65 70 75 80
Leu Glu Leu Gly Asp Ser Ala Leu Tyr Phe Cys Ala Ser Ser Val Gly
85 90 95
Asp Leu Asn Asn Glu Gln Phe Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu
115 120 125
Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys
130 135 140
Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val
145 150 155 160
Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro Leu
165 170 175
Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg
180 185 190
Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg
195 200 205
Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln
210 215 220
Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly
225 230 235 240
Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu
245 250 255
Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr
260 265 270
Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys
275 280 285
Asp Ser Arg Gly
290
<210> 991
<211> 311
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 52- бета нативный
<400> 991
Met Gly Phe Arg Leu Leu Cys Cys Val Ala Phe Cys Leu Leu Gly Ala
1 5 10 15
Gly Pro Val Asp Ser Gly Val Thr Gln Thr Pro Lys His Leu Ile Thr
20 25 30
Ala Thr Gly Gln Arg Val Thr Leu Arg Cys Ser Pro Arg Ser Gly Asp
35 40 45
Leu Ser Val Tyr Trp Tyr Gln Gln Ser Leu Asp Gln Gly Leu Gln Phe
50 55 60
Leu Ile Gln Tyr Tyr Asn Gly Glu Glu Arg Ala Lys Gly Asn Ile Leu
65 70 75 80
Glu Arg Phe Ser Ala Gln Gln Phe Pro Asp Leu His Ser Glu Leu Asn
85 90 95
Leu Ser Ser Leu Glu Leu Gly Asp Ser Ala Leu Tyr Phe Cys Ala Ser
100 105 110
Ser Val Gly Asp Leu Asn Asn Glu Gln Phe Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 992
<211> 311
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 52 - бета
цистеин-модифицированный
<400> 992
Met Gly Phe Arg Leu Leu Cys Cys Val Ala Phe Cys Leu Leu Gly Ala
1 5 10 15
Gly Pro Val Asp Ser Gly Val Thr Gln Thr Pro Lys His Leu Ile Thr
20 25 30
Ala Thr Gly Gln Arg Val Thr Leu Arg Cys Ser Pro Arg Ser Gly Asp
35 40 45
Leu Ser Val Tyr Trp Tyr Gln Gln Ser Leu Asp Gln Gly Leu Gln Phe
50 55 60
Leu Ile Gln Tyr Tyr Asn Gly Glu Glu Arg Ala Lys Gly Asn Ile Leu
65 70 75 80
Glu Arg Phe Ser Ala Gln Gln Phe Pro Asp Leu His Ser Glu Leu Asn
85 90 95
Leu Ser Ser Leu Glu Leu Gly Asp Ser Ala Leu Tyr Phe Cys Ala Ser
100 105 110
Ser Val Gly Asp Leu Asn Asn Glu Gln Phe Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 993
<211> 113
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 52 бета, вариабельная область
<400> 993
Asp Ser Gly Val Thr Gln Thr Pro Lys His Leu Ile Thr Ala Thr Gly
1 5 10 15
Gln Arg Val Thr Leu Arg Cys Ser Pro Arg Ser Gly Asp Leu Ser Val
20 25 30
Tyr Trp Tyr Gln Gln Ser Leu Asp Gln Gly Leu Gln Phe Leu Ile Gln
35 40 45
Tyr Tyr Asn Gly Glu Glu Arg Ala Lys Gly Asn Ile Leu Glu Arg Phe
50 55 60
Ser Ala Gln Gln Phe Pro Asp Leu His Ser Glu Leu Asn Leu Ser Ser
65 70 75 80
Leu Glu Leu Gly Asp Ser Ala Leu Tyr Phe Cys Ala Ser Ser Val Gly
85 90 95
Asp Leu Asn Asn Glu Gln Phe Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Leu
<210> 994
<211> 12
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 52 бета CDR3
<400> 994
Ala Ser Ser Val Gly Asp Leu Asn Asn Glu Gln Phe
1 5 10
<210> 995
<211> 256
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 53 - альфа нативный
<400> 995
Ala Gln Ser Val Ala Gln Pro Glu Asp Gln Val Asn Val Ala Glu Gly
1 5 10 15
Asn Pro Leu Thr Val Lys Cys Thr Tyr Ser Val Ser Gly Asn Pro Tyr
20 25 30
Leu Phe Trp Tyr Val Gln Tyr Pro Asn Arg Gly Leu Gln Phe Leu Leu
35 40 45
Lys Tyr Ile Thr Gly Asp Asn Leu Val Lys Gly Ser Tyr Gly Phe Glu
50 55 60
Ala Glu Phe Asn Lys Ser Gln Thr Ser Phe His Leu Lys Lys Pro Ser
65 70 75 80
Ala Leu Val Ser Asp Ser Ala Leu Tyr Phe Cys Ala Val Arg Asp Phe
85 90 95
Gly Ser Gly Thr Tyr Lys Tyr Ile Phe Gly Thr Gly Thr Arg Leu Lys
100 105 110
Val Leu Ala Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg
115 120 125
Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp
130 135 140
Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr
145 150 155 160
Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser
165 170 175
Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe
180 185 190
Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser
195 200 205
Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn
210 215 220
Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu
225 230 235 240
Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250 255
<210> 996
<211> 256
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 53 - альфа
цистеин-модифицированный
<400> 996
Ala Gln Ser Val Ala Gln Pro Glu Asp Gln Val Asn Val Ala Glu Gly
1 5 10 15
Asn Pro Leu Thr Val Lys Cys Thr Tyr Ser Val Ser Gly Asn Pro Tyr
20 25 30
Leu Phe Trp Tyr Val Gln Tyr Pro Asn Arg Gly Leu Gln Phe Leu Leu
35 40 45
Lys Tyr Ile Thr Gly Asp Asn Leu Val Lys Gly Ser Tyr Gly Phe Glu
50 55 60
Ala Glu Phe Asn Lys Ser Gln Thr Ser Phe His Leu Lys Lys Pro Ser
65 70 75 80
Ala Leu Val Ser Asp Ser Ala Leu Tyr Phe Cys Ala Val Arg Asp Phe
85 90 95
Gly Ser Gly Thr Tyr Lys Tyr Ile Phe Gly Thr Gly Thr Arg Leu Lys
100 105 110
Val Leu Ala Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg
115 120 125
Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp
130 135 140
Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr
145 150 155 160
Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser
165 170 175
Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe
180 185 190
Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser
195 200 205
Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn
210 215 220
Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu
225 230 235 240
Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250 255
<210> 997
<211> 275
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 53 - альфа нативный
<400> 997
Met Ala Ser Ala Pro Ile Ser Met Leu Ala Met Leu Phe Thr Leu Ser
1 5 10 15
Gly Leu Arg Ala Gln Ser Val Ala Gln Pro Glu Asp Gln Val Asn Val
20 25 30
Ala Glu Gly Asn Pro Leu Thr Val Lys Cys Thr Tyr Ser Val Ser Gly
35 40 45
Asn Pro Tyr Leu Phe Trp Tyr Val Gln Tyr Pro Asn Arg Gly Leu Gln
50 55 60
Phe Leu Leu Lys Tyr Ile Thr Gly Asp Asn Leu Val Lys Gly Ser Tyr
65 70 75 80
Gly Phe Glu Ala Glu Phe Asn Lys Ser Gln Thr Ser Phe His Leu Lys
85 90 95
Lys Pro Ser Ala Leu Val Ser Asp Ser Ala Leu Tyr Phe Cys Ala Val
100 105 110
Arg Asp Phe Gly Ser Gly Thr Tyr Lys Tyr Ile Phe Gly Thr Gly Thr
115 120 125
Arg Leu Lys Val Leu Ala Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr
130 135 140
Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr
145 150 155 160
Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val
165 170 175
Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys
180 185 190
Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala
195 200 205
Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser
210 215 220
Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr
225 230 235 240
Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile
245 250 255
Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu
260 265 270
Trp Ser Ser
275
<210> 998
<211> 275
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 53 - альфа
цистеин-модифицированный
<400> 998
Met Ala Ser Ala Pro Ile Ser Met Leu Ala Met Leu Phe Thr Leu Ser
1 5 10 15
Gly Leu Arg Ala Gln Ser Val Ala Gln Pro Glu Asp Gln Val Asn Val
20 25 30
Ala Glu Gly Asn Pro Leu Thr Val Lys Cys Thr Tyr Ser Val Ser Gly
35 40 45
Asn Pro Tyr Leu Phe Trp Tyr Val Gln Tyr Pro Asn Arg Gly Leu Gln
50 55 60
Phe Leu Leu Lys Tyr Ile Thr Gly Asp Asn Leu Val Lys Gly Ser Tyr
65 70 75 80
Gly Phe Glu Ala Glu Phe Asn Lys Ser Gln Thr Ser Phe His Leu Lys
85 90 95
Lys Pro Ser Ala Leu Val Ser Asp Ser Ala Leu Tyr Phe Cys Ala Val
100 105 110
Arg Asp Phe Gly Ser Gly Thr Tyr Lys Tyr Ile Phe Gly Thr Gly Thr
115 120 125
Arg Leu Lys Val Leu Ala Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr
130 135 140
Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr
145 150 155 160
Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val
165 170 175
Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys
180 185 190
Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala
195 200 205
Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser
210 215 220
Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr
225 230 235 240
Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile
245 250 255
Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu
260 265 270
Trp Ser Ser
275
<210> 999
<211> 115
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 53 альфа, вариабельная область
<400> 999
Ala Gln Ser Val Ala Gln Pro Glu Asp Gln Val Asn Val Ala Glu Gly
1 5 10 15
Asn Pro Leu Thr Val Lys Cys Thr Tyr Ser Val Ser Gly Asn Pro Tyr
20 25 30
Leu Phe Trp Tyr Val Gln Tyr Pro Asn Arg Gly Leu Gln Phe Leu Leu
35 40 45
Lys Tyr Ile Thr Gly Asp Asn Leu Val Lys Gly Ser Tyr Gly Phe Glu
50 55 60
Ala Glu Phe Asn Lys Ser Gln Thr Ser Phe His Leu Lys Lys Pro Ser
65 70 75 80
Ala Leu Val Ser Asp Ser Ala Leu Tyr Phe Cys Ala Val Arg Asp Phe
85 90 95
Gly Ser Gly Thr Tyr Lys Tyr Ile Phe Gly Thr Gly Thr Arg Leu Lys
100 105 110
Val Leu Ala
115
<210> 1000
<211> 6
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 53 альфа CDR1
<400> 1000
Val Ser Gly Asn Pro Tyr
1 5
<210> 1001
<211> 8
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 53 альфа CDR2
<400> 1001
Tyr Ile Thr Gly Asp Asn Leu Val
1 5
<210> 1002
<211> 13
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 53 альфа CDR3
<400> 1002
Ala Val Arg Asp Phe Gly Ser Gly Thr Tyr Lys Tyr Ile
1 5 10
<210> 1003
<211> 19
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 53 альфа CDR3
<400> 1003
Met Ala Ser Ala Pro Ile Ser Met Leu Ala Met Leu Phe Thr Leu Ser
1 5 10 15
Gly Leu Arg
<210> 1004
<211> 295
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 53 - бета нативный
<400> 1004
Gly Ala Gly Val Ser Gln Ser Pro Arg Tyr Lys Val Thr Lys Arg Gly
1 5 10 15
Gln Asp Val Ala Leu Arg Cys Asp Pro Ile Ser Gly His Val Ser Leu
20 25 30
Tyr Trp Tyr Arg Gln Ala Leu Gly Gln Gly Pro Glu Phe Leu Thr Tyr
35 40 45
Phe Asn Tyr Glu Ala Gln Gln Asp Lys Ser Gly Leu Pro Asn Asp Arg
50 55 60
Phe Ser Ala Glu Arg Pro Glu Gly Ser Ile Ser Thr Leu Thr Ile Gln
65 70 75 80
Arg Thr Glu Gln Arg Asp Ser Ala Met Tyr Arg Cys Ala Ser Ser Gly
85 90 95
Ser Gly Thr Ser Gly Tyr Asn Glu Gln Phe Phe Gly Pro Gly Thr Arg
100 105 110
Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
115 120 125
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
130 135 140
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
145 150 155 160
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
165 170 175
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
180 185 190
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
195 200 205
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
210 215 220
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
225 230 235 240
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
245 250 255
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
260 265 270
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
275 280 285
Lys Arg Lys Asp Ser Arg Gly
290 295
<210> 1005
<211> 295
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 53 - бета
цистеин-модифицированный
<400> 1005
Gly Ala Gly Val Ser Gln Ser Pro Arg Tyr Lys Val Thr Lys Arg Gly
1 5 10 15
Gln Asp Val Ala Leu Arg Cys Asp Pro Ile Ser Gly His Val Ser Leu
20 25 30
Tyr Trp Tyr Arg Gln Ala Leu Gly Gln Gly Pro Glu Phe Leu Thr Tyr
35 40 45
Phe Asn Tyr Glu Ala Gln Gln Asp Lys Ser Gly Leu Pro Asn Asp Arg
50 55 60
Phe Ser Ala Glu Arg Pro Glu Gly Ser Ile Ser Thr Leu Thr Ile Gln
65 70 75 80
Arg Thr Glu Gln Arg Asp Ser Ala Met Tyr Arg Cys Ala Ser Ser Gly
85 90 95
Ser Gly Thr Ser Gly Tyr Asn Glu Gln Phe Phe Gly Pro Gly Thr Arg
100 105 110
Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
115 120 125
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
130 135 140
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
145 150 155 160
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro
165 170 175
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
180 185 190
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
195 200 205
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
210 215 220
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
225 230 235 240
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
245 250 255
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
260 265 270
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
275 280 285
Lys Arg Lys Asp Ser Arg Gly
290 295
<210> 1006
<211> 314
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 53- бета нативный
<400> 1006
Met Gly Thr Ser Leu Leu Cys Trp Val Val Leu Gly Phe Leu Gly Thr
1 5 10 15
Asp His Thr Gly Ala Gly Val Ser Gln Ser Pro Arg Tyr Lys Val Thr
20 25 30
Lys Arg Gly Gln Asp Val Ala Leu Arg Cys Asp Pro Ile Ser Gly His
35 40 45
Val Ser Leu Tyr Trp Tyr Arg Gln Ala Leu Gly Gln Gly Pro Glu Phe
50 55 60
Leu Thr Tyr Phe Asn Tyr Glu Ala Gln Gln Asp Lys Ser Gly Leu Pro
65 70 75 80
Asn Asp Arg Phe Ser Ala Glu Arg Pro Glu Gly Ser Ile Ser Thr Leu
85 90 95
Thr Ile Gln Arg Thr Glu Gln Arg Asp Ser Ala Met Tyr Arg Cys Ala
100 105 110
Ser Ser Gly Ser Gly Thr Ser Gly Tyr Asn Glu Gln Phe Phe Gly Pro
115 120 125
Gly Thr Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro
130 135 140
Glu Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln
145 150 155 160
Lys Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val
165 170 175
Glu Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser
180 185 190
Thr Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg
195 200 205
Tyr Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn
210 215 220
Pro Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu
225 230 235 240
Asn Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val
245 250 255
Ser Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser
260 265 270
Tyr Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu
275 280 285
Gly Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met
290 295 300
Ala Met Val Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 1007
<211> 314
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 53 - бета
цистеин-модифицированный
<400> 1007
Met Gly Thr Ser Leu Leu Cys Trp Val Val Leu Gly Phe Leu Gly Thr
1 5 10 15
Asp His Thr Gly Ala Gly Val Ser Gln Ser Pro Arg Tyr Lys Val Thr
20 25 30
Lys Arg Gly Gln Asp Val Ala Leu Arg Cys Asp Pro Ile Ser Gly His
35 40 45
Val Ser Leu Tyr Trp Tyr Arg Gln Ala Leu Gly Gln Gly Pro Glu Phe
50 55 60
Leu Thr Tyr Phe Asn Tyr Glu Ala Gln Gln Asp Lys Ser Gly Leu Pro
65 70 75 80
Asn Asp Arg Phe Ser Ala Glu Arg Pro Glu Gly Ser Ile Ser Thr Leu
85 90 95
Thr Ile Gln Arg Thr Glu Gln Arg Asp Ser Ala Met Tyr Arg Cys Ala
100 105 110
Ser Ser Gly Ser Gly Thr Ser Gly Tyr Asn Glu Gln Phe Phe Gly Pro
115 120 125
Gly Thr Arg Leu Thr Val Leu Glu Asp Leu Lys Asn Val Phe Pro Pro
130 135 140
Glu Val Ala Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln
145 150 155 160
Lys Ala Thr Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val
165 170 175
Glu Leu Ser Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys
180 185 190
Thr Asp Pro Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg
195 200 205
Tyr Cys Leu Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn
210 215 220
Pro Arg Asn His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu
225 230 235 240
Asn Asp Glu Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val
245 250 255
Ser Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser
260 265 270
Tyr Gln Gln Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu
275 280 285
Gly Lys Ala Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met
290 295 300
Ala Met Val Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 1008
<211> 116
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 53 бета, вариабельная область
<400> 1008
Gly Ala Gly Val Ser Gln Ser Pro Arg Tyr Lys Val Thr Lys Arg Gly
1 5 10 15
Gln Asp Val Ala Leu Arg Cys Asp Pro Ile Ser Gly His Val Ser Leu
20 25 30
Tyr Trp Tyr Arg Gln Ala Leu Gly Gln Gly Pro Glu Phe Leu Thr Tyr
35 40 45
Phe Asn Tyr Glu Ala Gln Gln Asp Lys Ser Gly Leu Pro Asn Asp Arg
50 55 60
Phe Ser Ala Glu Arg Pro Glu Gly Ser Ile Ser Thr Leu Thr Ile Gln
65 70 75 80
Arg Thr Glu Gln Arg Asp Ser Ala Met Tyr Arg Cys Ala Ser Ser Gly
85 90 95
Ser Gly Thr Ser Gly Tyr Asn Glu Gln Phe Phe Gly Pro Gly Thr Arg
100 105 110
Leu Thr Val Leu
115
<210> 1009
<211> 6
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 53 бета CDR2
<400> 1009
Phe Asn Tyr Glu Ala Gln
1 5
<210> 1010
<211> 14
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 53 бета CDR3
<400> 1010
Ala Ser Ser Gly Ser Gly Thr Ser Gly Tyr Asn Glu Gln Phe
1 5 10
<210> 1011
<211> 19
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 53 бета, Сигнальный пептид
<400> 1011
Met Gly Thr Ser Leu Leu Cys Trp Val Val Leu Gly Phe Leu Gly Thr
1 5 10 15
Asp His Thr
<210> 1012
<211> 136
<212> Белок
<213> Mus musculus
<220>
<223> Мышиная константная область альфа
<400> 1012
Ile Gln Asn Pro Glu Pro Ala Val Tyr Gln Leu Lys Asp Pro Arg Ser
1 5 10 15
Gln Asp Ser Thr Leu Cys Leu Phe Thr Asp Phe Asp Ser Gln Ile Asn
20 25 30
Val Pro Lys Thr Met Glu Ser Gly Thr Phe Ile Thr Asp Lys Cys Val
35 40 45
Leu Asp Met Lys Ala Met Asp Ser Lys Ser Asn Gly Ala Ile Ala Trp
50 55 60
Ser Asn Gln Thr Ser Phe Thr Cys Gln Asp Ile Phe Lys Glu Thr Asn
65 70 75 80
Ala Thr Tyr Pro Ser Ser Asp Val Pro Cys Asp Ala Thr Leu Thr Glu
85 90 95
Lys Ser Phe Glu Thr Asp Met Asn Leu Asn Phe Gln Asn Leu Leu Val
100 105 110
Ile Val Leu Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu
115 120 125
Met Thr Leu Arg Leu Trp Ser Ser
130 135
<210> 1013
<211> 173
<212> Белок
<213> Mus musculus
<220>
<223> Мышиная константная область бета
<400> 1013
Glu Asp Leu Arg Asn Val Thr Pro Pro Lys Val Ser Leu Phe Glu Pro
1 5 10 15
Ser Lys Ala Glu Ile Ala Asn Lys Gln Lys Ala Thr Leu Val Cys Leu
20 25 30
Ala Arg Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp Trp Val Asn
35 40 45
Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Ala Tyr Lys
50 55 60
Glu Ser Asn Tyr Ser Tyr Cys Leu Ser Ser Arg Leu Arg Val Ser Ala
65 70 75 80
Thr Phe Trp His Asn Pro Arg Asn His Phe Arg Cys Gln Val Gln Phe
85 90 95
His Gly Leu Ser Glu Glu Asp Lys Trp Pro Glu Gly Ser Pro Lys Pro
100 105 110
Val Thr Gln Asn Ile Ser Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly
115 120 125
Ile Thr Ser Ala Ser Tyr Gln Gln Gly Val Leu Ser Ala Thr Ile Leu
130 135 140
Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr Ala Val Leu Val Ser
145 150 155 160
Thr Leu Val Val Met Ala Met Val Lys Arg Lys Asn Ser
165 170
<210> 1014
<211> 137
<212> Белок
<213> Mus musculus
<220>
<223> Мышиная константная область альфа
<400> 1014
Asn Ile Gln Asn Pro Glu Pro Ala Val Tyr Gln Leu Lys Asp Pro Arg
1 5 10 15
Ser Gln Asp Ser Thr Leu Cys Leu Phe Thr Asp Phe Asp Ser Gln Ile
20 25 30
Asn Val Pro Lys Thr Met Glu Ser Gly Thr Phe Ile Thr Asp Lys Thr
35 40 45
Val Leu Asp Met Lys Ala Met Asp Ser Lys Ser Asn Gly Ala Ile Ala
50 55 60
Trp Ser Asn Gln Thr Ser Phe Thr Cys Gln Asp Ile Phe Lys Glu Thr
65 70 75 80
Asn Ala Thr Tyr Pro Ser Ser Asp Val Pro Cys Asp Ala Thr Leu Thr
85 90 95
Glu Lys Ser Phe Glu Thr Asp Met Asn Leu Asn Phe Gln Asn Leu Ser
100 105 110
Val Met Gly Leu Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu
115 120 125
Leu Met Thr Leu Arg Leu Trp Ser Ser
130 135
<210> 1015
<211> 137
<212> Белок
<213> Mus musculus
<220>
<223> Мышиная константная область альфа
<220>
<221> ВАРИАНТ
<222> (48)...(48)
<223> X = неизвестный или другой
<400> 1015
Asn Ile Gln Asn Pro Glu Pro Ala Val Tyr Gln Leu Lys Asp Pro Arg
1 5 10 15
Ser Gln Asp Ser Thr Leu Cys Leu Phe Thr Asp Phe Asp Ser Gln Ile
20 25 30
Asn Val Pro Lys Thr Met Glu Ser Gly Thr Phe Ile Thr Asp Lys Xaa
35 40 45
Val Leu Asp Met Lys Ala Met Asp Ser Lys Ser Asn Gly Ala Ile Ala
50 55 60
Trp Ser Asn Gln Thr Ser Phe Thr Cys Gln Asp Ile Phe Lys Glu Thr
65 70 75 80
Asn Ala Thr Tyr Pro Ser Ser Asp Val Pro Cys Asp Ala Thr Leu Thr
85 90 95
Glu Lys Ser Phe Glu Thr Asp Met Asn Leu Asn Phe Gln Asn Leu Xaa
100 105 110
Val Xaa Xaa Leu Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu
115 120 125
Leu Met Thr Leu Arg Leu Trp Ser Ser
130 135
<210> 1016
<211> 173
<212> Белок
<213> Mus musculus
<220>
<223> Мышиная константная область бета
<220>
<221> ВАРИАНТ
<222> (57)...(57)
<223> X = неизвестный или другой
<400> 1016
Glu Asp Leu Arg Asn Val Thr Pro Pro Lys Val Ser Leu Phe Glu Pro
1 5 10 15
Ser Lys Ala Glu Ile Ala Asn Lys Gln Lys Ala Thr Leu Val Cys Leu
20 25 30
Ala Arg Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp Trp Val Asn
35 40 45
Gly Lys Glu Val His Ser Gly Val Xaa Thr Asp Pro Gln Ala Tyr Lys
50 55 60
Glu Ser Asn Tyr Ser Tyr Cys Leu Ser Ser Arg Leu Arg Val Ser Ala
65 70 75 80
Thr Phe Trp His Asn Pro Arg Asn His Phe Arg Cys Gln Val Gln Phe
85 90 95
His Gly Leu Ser Glu Glu Asp Lys Trp Pro Glu Gly Ser Pro Lys Pro
100 105 110
Val Thr Gln Asn Ile Ser Ala Glu Ala Trp Gly Arg Ala Asp Cys Gly
115 120 125
Ile Thr Ser Ala Ser Tyr Gln Gln Gly Val Leu Ser Ala Thr Ile Leu
130 135 140
Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr Ala Val Leu Val Ser
145 150 155 160
Thr Leu Val Val Met Ala Met Val Lys Arg Lys Asn Ser
165 170
<210> 1017
<211> 137
<212> Белок
<213> Mus musculus
<220>
<223> Мышиная константная область альфа
Цистеин-замещенная
<400> 1017
Asn Ile Gln Asn Pro Glu Pro Ala Val Tyr Gln Leu Lys Asp Pro Arg
1 5 10 15
Ser Gln Asp Ser Thr Leu Cys Leu Phe Thr Asp Phe Asp Ser Gln Ile
20 25 30
Asn Val Pro Lys Thr Met Glu Ser Gly Thr Phe Ile Thr Asp Lys Cys
35 40 45
Val Leu Asp Met Lys Ala Met Asp Ser Lys Ser Asn Gly Ala Ile Ala
50 55 60
Trp Ser Asn Gln Thr Ser Phe Thr Cys Gln Asp Ile Phe Lys Glu Thr
65 70 75 80
Asn Ala Thr Tyr Pro Ser Ser Asp Val Pro Cys Asp Ala Thr Leu Thr
85 90 95
Glu Lys Ser Phe Glu Thr Asp Met Asn Leu Asn Phe Gln Asn Leu Ser
100 105 110
Val Met Gly Leu Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu
115 120 125
Leu Met Thr Leu Arg Leu Trp Ser Ser
130 135
<210> 1018
<211> 137
<212> Белок
<213> Mus musculus
<220>
<223> Мышиная константная область альфа
Трансмембранно-модифицированная
<400> 1018
Asn Ile Gln Asn Pro Glu Pro Ala Val Tyr Gln Leu Lys Asp Pro Arg
1 5 10 15
Ser Gln Asp Ser Thr Leu Cys Leu Phe Thr Asp Phe Asp Ser Gln Ile
20 25 30
Asn Val Pro Lys Thr Met Glu Ser Gly Thr Phe Ile Thr Asp Lys Thr
35 40 45
Val Leu Asp Met Lys Ala Met Asp Ser Lys Ser Asn Gly Ala Ile Ala
50 55 60
Trp Ser Asn Gln Thr Ser Phe Thr Cys Gln Asp Ile Phe Lys Glu Thr
65 70 75 80
Asn Ala Thr Tyr Pro Ser Ser Asp Val Pro Cys Asp Ala Thr Leu Thr
85 90 95
Glu Lys Ser Phe Glu Thr Asp Met Asn Leu Asn Phe Gln Asn Leu Leu
100 105 110
Val Ile Val Leu Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu
115 120 125
Leu Met Thr Leu Arg Leu Trp Ser Ser
130 135
<210> 1019
<211> 498
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 17 - альфа нативный
<400> 1019
gagtaattct tctctcacca tgccaggttc acttcacagt acagagtcct gaaaataaag 60
aagaaaattt ttttttatct agaaaaagaa ccaaacatgt cactttctag cctgctgaag 120
gtggtcacag cttcactgtg gctaggacct ggcattgccc agaagataac tcaaacccaa 180
ccaggaatgt tcgtgcagga aaaggaggct gtgactctgg actgcacata tgacaccagt 240
gatccaagtt atggtctatt ctggtacaag cagcccagca gtggggaaat gatttttctt 300
atttatcagg ggtcttatga ccagcaaaat gcaacagaag gtcgctactc attgaatttc 360
cagaaggcaa gaaaatccgc caaccttgtc atctccgctt cacaactggg ggactcagca 420
atgtacttct gtgcaatgag agagggccgg ggtgacaaga tcatctttgg aaaagggaca 480
cgacttcata ttctcccc 498
<210> 1020
<211> 420
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 17 - бета нативный
<400> 1020
gattctttct tcaaagcagc catgggaatc aggctcctct gtcgtgtggc cttttgtttc 60
ctggctgtag gcctcgtaga tgtgaaagta acccagagct cgagatatct agtcaaaagg 120
acgggagaga aagtttttct ggaatgtgtc caggatatgg accatgaaaa tatgttctgg 180
tatcgacaag acccaggtct ggggctacgg ctgatctatt tctcatatga tgttaaaatg 240
aaagaaaaag gagatattcc tgaggggtac agtgtctcta gagagaagaa ggagcgcttc 300
tccctgattc tggagtccgc cagcaccaac cagacatcta tgtacctctg tgccagcagt 360
ttatgggggc gcagcaatca gccccagcat tttggtgatg ggactcgact ctccatccta 420
<210> 1021
<211> 481
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 18 - альфа нативный
<400> 1021
agagtgttta tcttgtgagc cattctccat atttcagata taagatttca gttctcagtg 60
agtctaagtg acagaaggaa tggagaccct cttgggcctg cttatccttt ggctgcagct 120
gcaatgggtg agcagcaaac aggaggtgac gcagattcct gcagctctga gtgtcccaga 180
aggagaaaac ttggttctca actgcagttt cactgatagc gctatttaca acctccagtg 240
gtttaggcag gaccctggga aaggtctcac atctctgttg cttattcagt caagtcagag 300
agagcaaaca agtggaagac ttaatgcctc gctggataaa tcatcaggac gtagtacttt 360
atacattgca gcttctcagc ctggtgactc agccacctac ctctgtgctg aggggaatgc 420
tggtggtact agctatggaa agctgacatt tggacaaggg accatcttga ctgtccatcc 480
a 481
<210> 1022
<211> 420
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 18 - бета нативный
<400> 1022
gattctttct tcaaagcagc catgggaatc aggctcctct gtcgtgtggc cttttgtttc 60
ctggctgtag gcctcgtaga tgtgaaagta acccagagct cgagatatct agtcaaaagg 120
acgggagaga aagtttttct ggaatgtgtc caggatatgg accatgaaaa tatgttctgg 180
tatcgacaag acccaggtct ggggctacgg ctgatctatt tctcatatga tgttaaaatg 240
aaagaaaaag gagatattcc tgaggggtac agtgtctcta gagagaagaa ggagcgcttc 300
tccctgattc tggagtccgc cagcaccaac cagacatcta tgtacctctg tgccagcagt 360
ttttgggggc gatctaattc acccctccac tttgggaacg ggaccaggct cactgtgaca 420
<210> 1023
<211> 501
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 19 - альфа нативный
<400> 1023
gaattcttct ctcaccatgc caggttcacc tcacagtaca gagtcctgaa aataaagaag 60
aagatttttt tttatctaga aaaggaacca aacatgtcac tttctagcct gctgaaggtg 120
gtcacagctt cactgtggct aggacctggc attgcccaga agataactca aacccaacca 180
ggaatgttcg tgcaggaaaa ggaggctgtg actctggact gcacatatga caccagtgat 240
caaagttatg gtctattctg gtacaagcag cccagcagtg gggaaatgat ttttcttatt 300
tatcaggggt cttatgacga gcaaaatgca acagaaggtc gctactcatt gaatttccag 360
aaggcaagaa aatccgccaa ccttgtcatc tccgcttcac aactggggga ctcagcaatg 420
tatttctgtg caatgagaga gaataccggc actgccagta aactcacctt tgggactgga 480
acaagacttc aggtcacgct c 501
<210> 1024
<211> 420
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 19 - бета нативный
<400> 1024
gattctttct tcaaagcagc catgggaatc aggctcctct gtcgtgtggc cttttgtttc 60
ctggctgtag gcctcgtaga tgtgaaagta acccagagct cgagatatct agtcaaaagg 120
acgggagaga aagtttttct ggaatgtgtc caggatatgg accatgaaaa tatgttctgg 180
tatcgacaag acccaggtct ggggctacgg ctgatctatt tctcatatga tgttaaaatg 240
aaagaaaaag gagatattcc tgaggggtac agtgtctcta gagagaagaa ggagcgcttc 300
tccctgattc tggagtccgc cagcaccaac cagacatcta tgtacctctg tgccagcagt 360
tcttggggac aaagcacagg cgagcagtac ttcgggccgg gcaccaggct cacggtcaca 420
<210> 1025
<211> 516
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 20 - альфа нативный
<400> 1025
gagaagcctc acacagccca gtaactttgc tagtacctct tgagtgcaag gtggagaatt 60
aagatctgga tttgagacgg agcacggaac atttcactca ggggaagagc tatgaacatg 120
ctgactgcca gcctgttgag ggcagtcata gcctccatct gtgttgtatc cagcatggct 180
cagaaggtaa ctcaagcgca gactgaaatt tctgtggtgg agaaggagga tgtgaccttg 240
gactgtgtgt atgaaacccg tgatactact tattacttat tctggtacaa gcaaccacca 300
agtggagaat tggttttcct tattcgtcgg aactcttttg atgagcaaaa tgaaataagt 360
ggtcggtatt cttggaactt ccagaaatcc accagttcct tcaacttcac catcacagcc 420
tcacaagtcg tggactcagc agtatacttc tgtgctctgt ggactggggc aaacaacctc 480
ttctttggga ctggaacgag actcaccgtt attccc 516
<210> 1026
<211> 436
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 20 - бета нативный
<400> 1026
gacaactctc cccagagaag gtggtgtgag gccatcacgg aagatgctgc tgcttctgct 60
gcttctgggg ccaggctccg ggcttggtgc tgtcgtctct caacatccga gcagggttat 120
ctgtaagagt ggaacctctg tgaagatcga gtgccgttcc ctggactttc aggccacaac 180
tatgttttgg tatcgtcagt tcccgaaaca gagtctcatg ctgatggcaa cttccaatga 240
gggctccaag gccacatacg agcaaggcgt cgagaaggac aagtttctca tcaaccatgc 300
aagcctgacc ttgtccactc tgacagtgac cagtgcccat cctgaagaca gcagcttcta 360
catctgcagt gctagctcgt tggcccgtcg ctcctacgag cagtacttcg ggccgggcac 420
caggctcacg gtcaca 436
<210> 1027
<211> 510
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 21 - альфа нативный
<400> 1027
gagtaattct tctctcacca tgccaggttc acctcacagt acagagtcct gaaaataaag 60
aagaagattt ttttttatct agaaaaggaa ccaaacatgt cactttctag cctgctgaag 120
gtggtcacag cttcactgtg gctaggacct ggcattgccc agaagataac tcaaacccaa 180
ccaggaatgt tcgtgcagga aaaggaggct gtgactctgg actgcacata tgacaccagt 240
gatcaaagtt atggtctatt ctggtacaag cagcccagca gtggggaaat gatttttctt 300
atttatcagg ggtcttatga cgagcaaaat gcaacagaag gtcgctactc attgaatttc 360
cagaaggcaa gaaaatccgc caaccttgtc atctccgctt cacaactggg ggactcagca 420
atgtatttct gtgcaatgag agagggccgg gtcacgggag gaggaaacaa actcaccttt 480
gggacaggca ctcagctaaa agtggaactc 510
<210> 1028
<211> 480
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 21 - бета нативный
<400> 1028
gagaatgacc cccttgagag tactgttccc ctatcaccga tgcacagacc cagaagaccc 60
ctccatcctg tagcacctgc catgagcatc gggctcctgt gctgtgtggc cttttctctc 120
ctgtgggcaa gtccagtgaa tgctggtgtc actcagaccc caaaattcca ggtcctgaag 180
acaggacaga gcatgacact gcagtgtgcc caggatatga accataactc catgtactgg 240
tatcgacaag acccaggcat gggactgagg ctgatttatt actcagcttc tgagggtacc 300
actgacaaag gagaagtccc caatggctac aatgtctcca gattaaacaa acgggagttc 360
tcgctcaggc tggagtcggc tgctccctcc cagacatctg tgtacttctg tgccagcagg 420
ccctggggga accagaacac tgaagctttc tttggacaag gcaccagact cacagttgta 480
<210> 1029
<211> 468
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 22 - альфа нативный
<400> 1029
acatgaagtg tctaccttct gcagactcca atggctcagg aactgggaat gcagtgccag 60
gctcgtggta tcctgcagca gatgtgggga gttttccttc tttatgtttc catgaagatg 120
ggaggcacta caggacaaaa cattgaccag cccactgaga tgacagctac ggaaggtgcc 180
attgtccaga tcaactgcac gtaccagaca tctgggttca acgggctgtt ctggtaccag 240
caacatgctg gcgaagcacc cacatttctg tcttacaatg ttctggatgg tttggaggag 300
aaaggtcgtt tttcttcatt ccttagtcgg tctaaagggt acagttacct ccttttgaag 360
gagctccaga tgaaagactc tgcctcttac ctctgtgctg tgcgcgcacg tatggatagc 420
aactatcagt taatctgggg cgctgggacc aagctaatta taaagcca 468
<210> 1030
<211> 444
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 22 - бета нативный
<400> 1030
gaggaggacc aagccctgaa tcaggtgcag tgctgcctgc cccactgtgc catgggccct 60
gggctcctct gctgggtgct gctttgtctc ctgggagcag gcccagtgga cgctggagtc 120
acccaaagtc ccacacacct gatcaaaacg agaggacagc acgtgactct gagatgctct 180
cctatctctg ggcacaagag tgtgtcctgg taccaacagg tcctgggtca ggggccccag 240
tttatctttc agtattatga gaaagaagag agaggaagag gaaacttccc tgatcgattc 300
tcagctcgcc agttccctaa ctatagctct gagctgaatg tgaacgcctt gttgctgggg 360
gactcggccc tgtatctctg tgccagcagc cgaactgaaa actatggcta caccttcggt 420
tcggggacca ggttaaccgt tgta 444
<210> 1031
<211> 501
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 23 - альфа нативный
<400> 1031
gaattcttct ctcaccatgc caggttcacc tcacagtaca gagtcctgaa aataaagaag 60
aagatttttt tttatctaga aaaggaacca aacatgtcac tttctagcct gctgaaggtg 120
gtcacagctt cactgtggct aggacctggc attgcccaga agataactca aacccaacca 180
ggaatgttcg tgcaggaaaa ggaggctgtg actctggact gcacatatga caccagtgat 240
caaagttatg gtctattctg gtacaagcag cccagcagtg gggaaatgat ttttcttatt 300
tatcaggggt cttatgacga gcaaaatgca acagaaggtc gctactcatt gaatttccag 360
aaggcaagaa aatccgccaa ccttgtcatc tccgcttcac aactggggga ctcagcaatg 420
tatttctgtg caatgagtcc tccgggtggt tctgcaaggc aactgacctt tggatctggg 480
acacaattga ctgttttacc t 501
<210> 1032
<211> 413
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 23 - бета нативный
<400> 1032
gcttcaaagc agccatggga atcaggctcc tctgtcgtgt ggccttttgt ttcctggctg 60
taggcctcgt agatgtgaaa gtaacccaga gctcgagata tctagtcaaa aggacgggag 120
agaaagtttt tctggaatgt gtccaggata tggaccatga aaatatgttc tggtatcgac 180
aagacccagg tctggggcta cggctgatct atttctcata tgatgttaaa atgaaagaaa 240
aaggagatat tcctgagggg tacagtgtct ctagagagaa gaaggagcgc ttctccctga 300
ttctggagtc cgccagcacc aaccagacat ctatgtacct ctgtgccagc agtccctggg 360
gacaaagcaa tcagccccag cattttggtg atgggactcg actctccatc cta 413
<210> 1033
<211> 504
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 24 - альфа нативный
<400> 1033
gaattcttct ctcaccatgc caggttcacc tcacagtaca gagtcctgaa aataaagaag 60
aagatttttt tttatctaga aaaggaacca aacatgtcac tttctagcct gctgaaggtg 120
gtcacagctt cactgtggct aggacctggc attgcccaga agataactca aacccaacca 180
ggaatgttcg tgcaggaaaa ggaggctgtg actctggact gcacatatga caccagtgat 240
caaagttatg gtctcttctg gtacaagcag cccagcagtg gggaaatgat ttttcttatt 300
tatcaggggt cttatgacga gcaaaatgca acagaaggtc gctactcatt gaatttccag 360
aaggcaagaa aatccgccaa ccttgtcatc tccgcttcac aactggggga ctcagcaatg 420
tatttctgtg caatgagaga gggcaggggt gacagctggg ggaaattgca gtttggagca 480
gggacccagg ttgtggtcac ccca 504
<210> 1034
<211> 420
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 24 - бета нативный
<400> 1034
gattctttct tcaaagcagc catgggaatc aggctcctct gtcgtgtggc cttttgtttc 60
ctggctgtag gcctcgtaga tgtgaaagta acccagagct cgagatatct agtcaaaagg 120
acgggagaga aagtttttct ggaatgtgtc caggatatgg accatgaaaa tatgttctgg 180
tatcgacaag acccaggtct ggggctacgg ctgatctatt tctcatatga tgttaaaatg 240
aaagaaaaag gagatattcc tgaggggtac agtgtctcta gagagaagaa ggagcgcttc 300
tccctgattc tggagtccgc cagcaccaac cagacatcta tgtacctctg tgccagcagc 360
cccttcggga ggggctccta cgagcagtac ttcgggccgg gcaccaggct cacggtcaca 420
<210> 1035
<211> 508
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 25 - альфа нативный
<400> 1035
agtaattctt ctctcaccat gccaggttca cctcacagta cagagtcctg aaaataaaga 60
agaagatttt ttttatctag aaaaggaacc aaacatgtca ctttctagcc tgctgaaggt 120
ggtcacagct tcactgtggc taggacctgg cattgcccag aagataactc aaacccaacc 180
aggaatgttc gtgcaggaaa aggaggctgt gactctggac tgcacatatg acaccagtga 240
tcaaagttat ggtctcttct ggtacaagca gcccagcagt ggggaaatga tttttcttat 300
ttatcagggg tcttatgacg agcaaaatgc aacagaaggt cgctactcat tgaatttcca 360
gaaggcaaga aaatccgcca accttgtcat ctccgcttca caactggggg actcagcaat 420
gtatttctgt gcaatgagag agggctcgct cacgggagga ggaaacaaac tcacctttgg 480
gacaggcact cagctaaaag tggaactc 508
<210> 1036
<211> 422
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 25 - бета нативный
<400> 1036
gctattcttt cttcaaagca gccatgggaa tcaggctcct ctgtcgtgtg gccttttgtt 60
tcctggctgt aggcctcgta gatgtgaaag taacccagag ctcgagatat ctagtcaaaa 120
ggacgggaga gaaagttttt ctggaatgtg tccaggatat ggaccatgaa aatatgttct 180
ggtatcgaca agacccaggt ctggggctac ggctgatcta tttctcatat gatgttaaaa 240
tgaaagaaaa aggagatatt cctgaggggt acagtgtctc tagagagaag aaggagcgct 300
tctccctgat tctggagtcc gccagcacca accagacatc tatgtacctc tgtgccagca 360
gtttatgggg acagagcaat cagccccagc attttggtga tgggactcga ctctccatcc 420
ta 422
<210> 1037
<211> 498
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 26 - альфа нативный
<400> 1037
gagtaattct tctctcacca tgccaggttc acctcacagt acagagtcct gaaaataaag 60
aagaagattt ttttttatct agaaaaggaa ccaaacatgt cactttctag cctgctgaag 120
gtggtcacag cttcactgtg gctaggacct ggcattgccc agaagataac tcaaacccaa 180
ccaggaatgt tcgtgcagga aaaggaggct gtgactctgg actgcacata tgacaccagt 240
gatcaaagtt atggtctctt ctggtacaag cagcccagca gtggggaaat gatttttctt 300
atttatcagg ggtcttatga cgagcaaaat gcaacagaag gtcgctactc attgaatttc 360
cagaaggcaa gaaaatccgc caaccttgtc atctccgctt cacaactggg ggactcagca 420
atgtatttct gtgcaatgag agacgcccgg aacaatgaca tgcgctttgg agcagggacc 480
agactgacag taaaacca 498
<210> 1038
<211> 437
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 26 - бета нативный
<400> 1038
acagacccag aggacccctc catcctgcag ttcctgccat gagcctcggg ctcctgtgct 60
gtggggcctt ttctctcctg tgggcaggtc cagtgaatgc tggtgtcact cagaccccaa 120
aattccgggt cctgaagaca ggacagagca tgacactgct gtgtgcccag gatatgaacc 180
atgaatacat gtactggtat cgacaagacc caggcatggg gctgaggctg attcattact 240
cagttggtga gggtacaact gccaaaggag aggtccctga tggctacaat gtctccagat 300
taaaaaaaca gaatttcctg ctggggttgg agtcggctgc tccctcccaa acatctgtgt 360
acttctgtgc cagcagttac tatgctagcg ggagaaacta cgagcagtac ttcgggccgg 420
gcaccaggct cacggtc 437
<210> 1039
<211> 410
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 27 - альфа нативный
<400> 1039
gaatgtgcac ttgaacaatg aagttggtga caagcattac tgtactccta tctttgggta 60
ttatgggtga tgctaagacc acacagccaa attcaatgga gagtaacgaa gaagagcctg 120
ttcacttgcc ttgtaaccac tccacaatca gtggaactga ttacatacat tggtatcgac 180
agcttccctc ccagggtcca gagtacgtga ttcatggtct tacaagcaat gtgaacaaca 240
gaatggcctc tctggcaatc gctgaagaca gaaagtccag taccttgatc ctgcaccgtg 300
ctaccttgag agatgctgct gtgtactact gcatcctgag agtccccccc cagtctgggg 360
gttaccagaa agttaccttt ggaactggaa caaagctcca agtcatccca 410
<210> 1040
<211> 480
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 27 - бета нативный
<400> 1040
gagaatgacc cccttgagag tactgttccc ctatcaccga tgcacagacc cagaagaccc 60
ctccatcctg tagcacctgc catgagcatc gggctcctgt gctgtgtggc cttttctctc 120
ctgtgggcaa gtccagtgaa tgctggtgtc actcagaccc caaaattcca ggtcctgaag 180
acaggacaga gcatgacact gcagtgtgcc caggatatga accataactc catgtactgg 240
tatcgacaag acccaggcat gggactgagg ctgatttatt actcagcttc tgagggtacc 300
actgacaaag gagaagtccc caatggctac aatgtctcca gattaaacaa acgggagttc 360
tcgctcaggc tggagtcggc tgctccctcc cagacatctg tgtacttctg tgccagcagt 420
gaatttggga gcttgaatga aaaactgttt tttggcagtg gaacccagct ctctgtcttg 480
<210> 1041
<211> 513
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 28 - альфа нативный
<400> 1041
aggacagatt ctttttatga ttcctaaagt ggaagaaata aagtatctct gctatgttca 60
tttctttttg gattgaaaat tttaatcctc agtgaaccag ggcagaaaag aatgatgata 120
tccttgagag ttttactggt gatcctgtgg cttcagttaa gctgggtttg gagccaacgg 180
aaggaggtgg agcaggatcc tggacccttc aatgttccag agggagccac tgtcgctttc 240
aactgtactt acagcaacag tgcttctcag tctttcttct ggtacagaca ggattgcagg 300
aaagaaccta agttgctgat gtccgtatac tccagtggta atgaagatgg aaggtttaca 360
gcacagctca atagagccag ccagtatatt tccctgctca tcagagactc caagctcagt 420
gattcagcca cctacctctg tgtggtgagg gggggtggta ctagctatgg aaagctgaca 480
tttggacaag ggaccatctt gactgtccat cca 513
<210> 1042
<211> 427
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 28 - бета нативный
<400> 1042
gacctattct ttcttcaaag cagccatggg aatcaggctc ctctgtcgtg tggccttttg 60
tttcctggct gtaggcctcg tagatgtgaa agtaacccag agctcgagat atctagtcaa 120
aaggacggga gagaaagttt ttctggaatg tgtccaggat atggaccatg aaaatatgtt 180
ctggtatcga caagacccag gtctggggct acggctgatc tatttctcat atgatgttaa 240
aatgaaagaa aaaggagata ttcctgaggg gtacagtgtc tctagagaga agaaggagcg 300
cttctccctg attctggagt ccgccagcac caaccagaca tctatgtacc tctgtgccag 360
cagcttgtgg ggcaggagct ctggaaacac catatatttt ggagagggaa gttggctcac 420
tgttgta 427
<210> 1043
<211> 493
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 29 - альфа нативный
<400> 1043
gtctcaccat gccaggttca cctcacagta cagagtcctg aaaataaaga agaagatttt 60
tttttatcta gaaaaggaac caaacatgtc actttctagc ctgctgaagg tggtcacagc 120
ttcactgtgg ctaggacctg gcattgccca gaagataact caaacccaac caggaatgtt 180
cgtgcaggaa aaggaggctg tgactctgga ctgcacatat gacaccagtg atcaaagtta 240
tggtctattt tggtacaagc agcccagcag tggggaaatg atttttctta tttatcaggg 300
gtcttatgac gagcaaaatg caacagaagg tcgctactca ttgaatttcc agaaggcaag 360
aaaatccgcc aaccttgtca tctccgcttc acaactgggg gactcagcaa tgtatttctg 420
tgcaatgaga ctaaataccg gcactgccag taaactcacc tttgggactg gaacaagact 480
tcaggtcacg ctc 493
<210> 1044
<211> 514
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 29 - бета нативный
<400> 1044
attccagcct ctgacaaact ctgcacatta ggccaggaga agcccccgag ccaagtctct 60
tttctcattc tcttccaaca agtgcttgga gctccaagaa ggcccccttt gcactatgag 120
caaccaggtg ctctgctgtg tggtcctttg tctcctggga gcaaacaccg tggatggtgg 180
aatcactcag tccccaaagt acctgttcag aaaggaagga cagaatgtga ccctgagttg 240
tgaacagaat ttgaaccacg atgccatgta ctggtaccga caggacccag ggcaagggct 300
gagattgatc tactactcac agatagtaaa tgactttcag aaaggagata tagctgaagg 360
gtacagcgtc tctcgggaga agaaggaatc ctttcctctc actgtgacat cggcccaaaa 420
gaacccgaca gctttctatc tctgtgccag tagtccctgg gggcgggcaa ccaatgagca 480
gttcttcggg ccagggacac ggctcaccgt gcta 514
<210> 1045
<211> 490
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 30 - альфа нативный
<400> 1045
gagtagctgt tgcttctgtt ctctggaaat tctttaaacc tagaatcaga cacaaaaact 60
gaactctggg tccacaatcc tcatttgtcc ttgaagtatg aggctggtgg caagagtaac 120
tgtgtttctg acctttggaa ctataattga tgctaagacc acccagcccc cctccatgga 180
ttgcgctgaa ggaagagctg caaacctgcc ttgtaatcac tctaccatca gtggaaatga 240
gtatgtgtat tggtatcgac agattcactc ccaggggcca cagtatatca ttcatggtct 300
aaaaaacaat gaaaccaatg aaatggcctc tctgatcatc acagaagaca gaaagtccag 360
caccttgatc ctgccccacg ctacgctgag agacactgct gtgtactatt gcatcgtcag 420
aggcacctct gtcttgcagg gaaatgagaa attaaccttt gggactggaa caagactcac 480
catcataccc 490
<210> 1046
<211> 456
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 30 - бета нативный
<400> 1046
gacaggacca gatgcctgag ctaggaaagg cctcattcct gctgtgatcc tgccatggat 60
acctggctcg tatgctgggc aatttttagt ctcttgaaag caggactcac agaacctgaa 120
gtcacccaga ctcccagcca tcaggtcaca cagatgggac aggaagtgat cttgcgctgt 180
gtccccatct ctaatcactt atacttctat tggtacagac aaatcttggg gcagaaagtc 240
gagtttctgg tttcctttta taataatgaa atctcagaga agtctgaaat attcgatgat 300
caattctcag ttgaaaggcc tgatggatca aatttcactc tgaagatccg gtccacaaag 360
ctggaggact cagccatgta cttctgtgcc agcagtcgac agcccagctc tggaaacacc 420
atatattttg gagagggaag ttggctcact gttgta 456
<210> 1047
<211> 1489
<212> ДНК
<213> Homo sapiens
<220>
<223> Человеческий TCR бета, константная область 2 (TRBC2)
<300>
<308> NCBI NG_001333.2
<309> 2016-08-31
<400> 1047
aggacctgaa aaacgtgttc ccacccgagg tcgctgtgtt tgagccatca gaagcagaga 60
tctcccacac ccaaaaggcc acactggtat gcctggccac aggcttctac cccgaccacg 120
tggagctgag ctggtgggtg aatgggaagg aggtgcacag tggggtcagc acagacccgc 180
agcccctcaa ggagcagccc gccctcaatg actccagata ctgcctgagc agccgcctga 240
gggtctcggc caccttctgg cagaaccccc gcaaccactt ccgctgtcaa gtccagttct 300
acgggctctc ggagaatgac gagtggaccc aggatagggc caaacccgtc acccagatcg 360
tcagcgccga ggcctggggt agagcaggtg agtggggcct ggggagatgc ctggaggaga 420
ttaggtgaga ccagctacca gggaaaatgg aaagatccag gtagcggaca agactagatc 480
cagaagaaag ccagagtgga caaggtggga tgatcaaggt tcacagggtc agcaaagcac 540
ggtgtgcact tcccccacca agaagcatag aggctgaatg gagcacctca agctcattct 600
tccttcagat cctgacacct tagagctaag ctttcaagtc tccctgagga ccagccatac 660
agctcagcat ctgagtggtg tgcatcccat tctcttctgg ggtcctggtt tcctaagatc 720
atagtgacca cttcgctggc actggagcag catgagggag acagaaccag ggctatcaaa 780
ggaggctgac tttgtactat ctgatatgca tgtgtttgtg gcctgtgagt ctgtgatgta 840
aggctcaatg tccttacaaa gcagcattct ctcatccatt tttcttcccc tgttttcttt 900
cagactgtgg cttcacctcc ggtaagtgag tctctccttt ttctctctat ctttcgccgt 960
ctctgctctc gaaccagggc atggagaatc cacggacaca ggggcgtgag ggaggccaga 1020
gccacctgtg cacaggtgcc tacatgctct gttcttgtca acagagtctt accagcaagg 1080
ggtcctgtct gccaccatcc tctatgagat cttgctaggg aaggccacct tgtatgccgt 1140
gctggtcagt gccctcgtgc tgatggccat ggtaaggagg agggtgggat agggcagatg 1200
atgggggcag gggatggaac atcacacatg ggcataaagg aatctcagag ccagagcaca 1260
gcctaatata tcctatcacc tcaatgaaac cataatgaag ccagactggg gagaaaatgc 1320
agggaatatc acagaatgca tcatgggagg atggagacaa ccagcgagcc ctactcaaat 1380
taggcctcag agcccgcctc ccctgcccta ctcctgctgt gccatagccc ctgaaaccct 1440
gaaaatgttc tctcttccac aggtcaagag aaaggattcc agaggctag 1489
<210> 1048
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRAC нацеливающий домен гРНК
<400> 1048
gagaaucaaa aucggugaau 20
<210> 1049
<211> 467
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 32 - альфа нативный
<400> 1049
gagcccagag aagtctgttc caggacgggc tctttcagga gcagctaaag tcaggggcca 60
tgtccaccat gtgatagaaa gacaagatgg tcctgaaatt ctccgtgtcc attctttgga 120
ttcagttggc atgggtgagc acccagctgc tggagcagag ccctcagttt ctaagcatcc 180
aagagggaga aaatctcact gtgtactgca actcctcaag tgttttttcc agcttacaat 240
ggtacagaca ggagcctggg gaaggtcctg tcctcctggt gacagtagtt acgggtggag 300
aagtgaagaa gctgaagaga ctaacctttc agtttggtga tgcaagaaag gacagttctc 360
tccacatcac tgcagcccag cctggtgata caggcctcta cctctgtgca ggaggtagca 420
actataaact gacatttgga aaaggaactc tcttaaccgt gaatcca 467
<210> 1050
<211> 447
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 32- бета нативный
<400> 1050
gagaatgctt actacagaga caccagcccc aagctaggag atcctgccat gggcttcagg 60
ctcctctgct gtgtggcctt ttgtctcctg ggagcaggcc cagtggattc tggagtcaca 120
caaaccccaa agcacctgat cacagcaact ggacagcgag tgacgctgag atgctcccct 180
aggtctggag acctctctgt gtactggtac caacagagcc tggaccaggg cctccagttc 240
ctcattcagt attataatgg agaagagaga gcaaaaggaa acattcttga acgattctcc 300
gcacaacagt tccctgactt gcactctgaa ctaaacctga gctctctgga gctgggggac 360
tcagctttgt atttctgtgc cagcagcgta gggggggatc attctgatga gcagttcttc 420
gggccaggga cacggctcac cgtgcta 447
<210> 1051
<211> 508
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 33 - альфа нативный
<400> 1051
gaaagcagat tctttttatg atttttaaag tagaaatatc cattctaggt gcatttttta 60
agggtttaaa atttgaatcc tcagtgaacc agggcagaga agaatgatga aatccttgag 120
agttttacta gtgatcctgt ggcttcagtt gagctgggtt tggagccaac agaaggaggt 180
ggagcagaat tctggacccc tcagtgttcc agagggagcc attgcctctc tcaactgcac 240
ttacagtgac cgaggttccc agtccttctt ctggtacaga caatattctg ggaaaagccc 300
tgagttgata atgttcatat actccaatgg tgacaaagaa gatggaaggt ttacagcaca 360
gctcaataaa gccagccagt atgtttctct gctcatcaga gactcccagc ccagtgattc 420
agccacctac ctctgtgccg tgaacgcgca ccatactgga ggcttcaaaa ctatctttgg 480
agcaggaaca agactatttg ttaaagca 508
<210> 1052
<211> 417
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 33 - бета нативный
<400> 1052
gattcctgct gtgatcctgc catggatacc tggctcgtat gctgggcaat ttttagtctc 60
ttgaaagcag gactcacaga acctgaagtc acccagactc ccagccatca ggtcacacag 120
atgggacagg aagtgatctt gcgctgtgtc cccatctcta atcacttata cttctattgg 180
tacagacaaa tcttggggca gaaagtcgag tttctggttt ccttttataa taatgaaatc 240
tcagagaagt ctgaaatatt cgatgatcaa ttctcagttg aaaggcctga tggatcaaat 300
ttcactctga agatccggtc cacaaagctg gaggactcag ccatgtactt ctgtgccagc 360
accccacggg acacctacga gcagtacttc gggccgggca ccaggctcac ggtcaca 417
<210> 1053
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC нацеливающий домен гРНК
<400> 1053
ggccucggcg cugacgaucu 20
<210> 1054
<211> 20
<212> ДНК
<213> Homo sapiens
<220>
<223> TRBC последовательность-мишень
<400> 1054
agatcgtcag cgccgaggcc 20
<210> 1055
<211> 397
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 35 - альфа нативный
<400> 1055
gattttggcc atggctttgc agagcactct gggggcggtg tggctagggc ttctcctcaa 60
ctctctctgg aaggttgcag aaagcaagga ccaagtgttt cagccttcca cagtggcatc 120
ttcagaggga gctgtggtgg aaatcttctg taatcactct gtgtccaatg cttacaactt 180
cttctggtac cttcacttcc cgggatgtgc accaagactc cttgttaaag gctcaaagcc 240
ttctcagcag ggacgataca acatgaccta tgaacggttc tcttcatcgc tgctcatcct 300
ccaggtgcgg gaggcagatg ctgctgttta ctactgtgct gtggcgggcg actacaagct 360
cagctttgga gccggaacca cagtaactgt aagagca 397
<210> 1056
<211> 450
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 35 - бета нативный
<400> 1056
gagaatgctt actacagaga caccagcccc aagctaggag atcctgccat gggcttcagg 60
ctcctctgct gtgtggcctt ttgtctcctg ggagcaggcc cagtggattc tggagtcaca 120
caaaccccaa agcacctgat cacagcaact ggacagcgag tgacgctgag atgctcccct 180
aggtctggag acctctctgt gtactggtac caacagagcc tggaccaggg cctccagttc 240
ctcattcagt attataatgg agaagagaga gcaaaaggaa acattcttga acgattctcc 300
gcacaacagt tccctgactt gcactctgaa ctaaacctga gctctctgga gctgggggac 360
tcagctttgt atttctgtgc cagtacaact agcggggatt cctcctacaa tgagcagttc 420
ttcgggccag ggacacggct caccgtgcta 450
<210> 1057
<211> 511
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 36 - альфа нативный
<400> 1057
gaggacagat tctttttatg attcctaaag tggaagaaat aaagtatctc tgctatgttc 60
atttcttttt ggattgaaaa ttttaatcct cagtgaacca gggcagaaaa gaatgatgat 120
atccttgaga gttttactgg tgatcctgtg gcttcagtta agctgggttt ggagccaacg 180
gaaggaggtg gagcaggatc ctggaccctt caatgttcca gagggagcca ctgtcgcttt 240
caactgtact tacagcaaca gtgcttctca gtctttcttc tggtacagac aggattgcag 300
gaaagaacct aagttgctga tgtccgtata ctccagtggt aatgaagatg gaaggtttac 360
agcacagctc aatagagcca gccagtatat ttccctgctc atcagagact ccaagctcag 420
tgattcagcc acctacctct gtgtggtgaa ccgggataac tatggtcaga attttgtctt 480
tggtcccgga accagattgt ccgtgctgcc c 511
<210> 1058
<211> 416
<212>
ДНК
<213> Homo sapiens
<220>
<223> TCR 36 - бета нативный
<400> 1058
attcctgctg tgatcctgcc atggatacct ggctcgtatg ctgggcaatt tttagtctct 60
tgaaagcagg actcacagaa cctgaagtca cccagactcc cagccatcag gtcacacaga 120
tgggacagga agtgatcttg cgctgtgtcc ccatctctaa tcacttatac ttctattggt 180
acagacaaat cttggggcag aaagtcgagt ttctggtttc cttttataat aatgaaatct 240
cagagaagtc tgaaatattc gatgatcaat tctcagttga aaggcctgat ggatcaaatt 300
tcactctgaa gatccggtcc acaaagctgg aggactcagc catgtacttc tgtgccatga 360
ccggacggag caactacgag cagtacttcg ggccgggcac caggctcacg gtcaca 416
<210> 1059
<211> 464
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 37 - альфа нативный
<400> 1059
gatatttgga tagccctggc caactttcaa ggctcctaaa tctgagtttt cagtgaactg 60
gacagaaaaa aaaaatgaag aagctactag caatgattct gtggcttcaa ctagaccggt 120
taagtggaga gctgaaagtg gaacaaaacc ctctgttcct gagcatgcag gagggaaaaa 180
actataccat ctactgcaat tattcaacca cttcagacag actgtattgg tacaggcagg 240
atcctgggaa aagtctggaa tctctgtttg tgttgctatc aaatggagca gtgaagcagg 300
agggacgatt aatggcctca cttgatacca aagcccgtct cagcaccctc cacatcacag 360
ctgccgtgca tgacctctct gccacctact tctgtgccgg gtactctggg gctgggagtt 420
accaactcac tttcgggaag gggaccaaac tctcggtcat acca 464
<210> 1060
<211> 447
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 37 - бета нативный
<400> 1060
gacagtgaca ctgatctggt aaagccccca tcctggcctg accctgccat gggcaccagg 60
ctcctctgct gggtggtcct gggtttccta gggacagatc acacaggtgc tggagtctcc 120
cagtccccta ggtacaaagt cgcaaagaga ggacaggatg tagctctcag gtgtgatcca 180
atttcgggtc atgtatccct tttttggtac caacaggccc tggggcaggg gccagagttt 240
ctgacttatt tccagaatga agctcaacta gacaaatcgg ggctgcccag tgatcgcttc 300
tttgcagaaa ggcctgaggg atccgtctcc actctgaaga tccagcgcac acagcaggag 360
gactccgccg tgtatctctg tgccagcagc ttacttctgg gggcctatgg ctacaccttc 420
ggttcgggga ccaggttaac cgttgta 447
<210> 1061
<211> 513
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 38 - альфа нативный
<400> 1061
gaggctttgt cgggtggagc tgattggttg caggagcagc aacagttcca gagccaagtc 60
atgacaccga cctccccaag gtttagttaa atatatctta tggtgaaaat gcccggagca 120
agaaggcaaa gcatcatgaa gaggatattg ggagctctgc tggggctctt gagtgcccag 180
gtttgctgtg tgagaggaat acaagtggag cagagtcctc cagacctgat tctccaggag 240
ggagccaatt ccacgctgcg gtgcaatttt tctgactctg tgaacaattt gcagtggttt 300
catcaaaacc cttggggaca gctcatcaac ctgttttaca ttccctcagg gacaaaacag 360
aatggaagat taagcgccac gactgtcgct acggaacgct acagcttatt gtacatttcc 420
tcttcccaga ccacagactc aggcgtttat ttctgtgctg tggggttcaa tgacatgcgc 480
tttggagcag ggaccagact gacagtaaaa cca 513
<210> 1062
<211> 426
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 38 - бета нативный
<400> 1062
gaggtgcagt gctgcctgcc ccactgtgcc atgggccctg ggctcctctg ctgggtgctg 60
ctttgtctcc tgggagcagg ctcagtggag actggagtca cccaaagtcc cacacacctg 120
atcaaaacga gaggacagca agtgactctg agatgctctt ctcagtctgg gcacaacact 180
gtgtcctggt accaacaggc cctgggtcag gggccccagt ttatctttca gtattatagg 240
gaggaagaga atggcagagg aaacttccct cctagattct caggtctcca gttccctaat 300
tatagctctg agctgaatgt gaacgccttg gagctggacg actcggccct gtatctctgt 360
gccagcagct tggtagcggg tggagagacc cagtacttcg ggccaggcac gcggctcctg 420
gtgctc 426
<210> 1063
<211> 371
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 39 - альфа нативный
<400> 1063
gaataatctt gtggatgcag ctgacatggg tcagtggtca acagctgaat cagagtcctc 60
aatctatgtt tatccaggaa ggagaagatg tctccatgaa ctgcacttct tcaagcatat 120
ttaacacctg gctatggtac aagcaggaac ctggggaagg tcctgtcctc ttgatagcct 180
tatataaggc tggtgaattg acctcaaatg gaagactgac tgctcagttt ggtataacca 240
gaaaggacag cttcctgaat atctcagcat ccatacctag tgatgtaggc atctacttct 300
gtgctggata ctcatcttct aacgactaca agctcagctt tggagccgga accacagtaa 360
ctgtaagagc a 371
<210> 1064
<211> 420
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 39 - бета нативный
<400> 1064
gattcctgct gtgatcctgc catggatacc tggctcgtat gctgggcaat ttttagtctc 60
ttgaaagcag gactcacaga acctgaagtc acccagactc ccagccatca ggtcacacag 120
atgggacagg aagtgatctt gcgctgtgtc cccatctcta atcacttata cttctattgg 180
tacagacaaa tcttggggca gaaagtcgag tttctggttt ccttttataa taatgaaatc 240
tcagagaagt ctgaaatatt cgatgatcaa ttctcagttg aaaggcctga tggatcaaat 300
ttcactctga agatccggtc cacaaagctg gaggactcag ccatgtactt ctgtgccagc 360
acccctaggg acaggggcaa ggaagctttc tttggacaag gcaccagact cacagttgta 420
<210> 1065
<211> 502
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 40 - альфа нативный
<400> 1065
gctttccaaa atgcacacat ctactttcca aaatagaccg caactatttc tattgatctg 60
gaagaagctt gtaccaggca acccatttag gagaagttgg atgaagaggg agagggagat 120
gctactcatc acatcaatgt tggtcttatg gatgcaattg tcacaggtga atggacaaca 180
ggtaatgcaa attcctcagt accagcatgt acaagaagga gaggacttca ccacgtactg 240
caattcctca actactttaa gcaatataca gtggtataag caaaggcctg gtggacatcc 300
cgtttttttg atacagttag tgaagagtgg agaagtgaag aagcagaaaa gactgacatt 360
tcagtttgga gaagcaaaaa agaacagctc cctgcacatc acagccaccc agactacaga 420
tgtaggaacc tacttctgtg cagggcggaa caacttcaac aaattttact ttggatctgg 480
gaccaaactc aatgtaaaac ca 502
<210> 1066
<211> 417
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 40 - бета нативный
<400> 1066
gattcctgct gtgatcctgc catggatacc tggctcgtat gctgggcaat ttttagtctc 60
ttgaaagcag gactcacaga acctgaagtc acccagactc ccagccatca ggtcacacag 120
atgggacagg aagtgatctt gcgctgtgtc cccatctcta atcacttata cttctattgg 180
tacagacaaa tcttggggca gaaagtcgag tttctggttt ccttttataa taatgaaatc 240
tcagagaagt ctgaaatatt cgatgatcaa ttctcagttg aaaggcctga tggatcaaat 300
ttcactctga agatccggtc cacaaagctg gaggactcag ccatgtactt ctgtgccata 360
acggcccgga gctcctacga gcagtacttc gggccgggca ccaggctcac ggtcaca 417
<210> 1067
<211> 451
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 41 - альфа нативный
<400> 1067
gctttttgga ttgaaaattt taatcctcag tgaaccaggg cagaaaagaa tgatgatatc 60
cttgagagtt ttactggtga tcctgtggct tcagttaagc tgggtttgga gccaacggaa 120
ggaggtggag caggatcctg gacccttcaa tgttccagag ggagccactg tcgctttcaa 180
ctgtacttac agcaacagtg cttctcagtc tttcttctgg tacagacagg attgcaggaa 240
agaacctaag ttgctgatgt ccgtatactc cagtggtaat gaagatggaa ggtttacagc 300
acagctcaat agagccagcc agtatatttc cctgctcatc agagactcca agctcagtga 360
ttcagccacc tacctctgtg tggtgacctt cgcccttact ggaggcttca aaactatctt 420
tggagcagga acaagactat ttgttaaagc a 451
<210> 1068
<211> 426
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 41 - бета нативный
<400> 1068
gctcattcct gctgtgatcc tgccatggat acctggctcg tatgctgggc aatttttagt 60
ctcttgaaag caggactcac agaacctgaa gtcacccaga ctcccagcca tcaggtcaca 120
cagatgggac aggaagtgat cttgcgctgt gtccccatct ctaatcactt atacttctat 180
tggtacagac aaatcttggg gcagaaagtc gagtttctgg tttcctttta taataatgaa 240
atctcagaga agtctgaaat attcgatgat caattctcag ttgaaaggcc tgatggatca 300
aatttcactc tgaagatccg gtccacaaag ctggaggact cagccatgta cttctgtgcc 360
agcaatcccc gggacagggt ctcctacgag cagtacttcg ggccgggcac caggctcacg 420
gtcaca 426
<210> 1069
<211> 502
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 42- альфа нативный
<400> 1069
gctttccaaa atgcacacat ctactttcca aaatagaccg caactatttc tattgatctg 60
gaagaagctt gtaccaggca acccatttag gagaagttgg atgaagaggg agagggagat 120
gctactcatc acatcaatgt tggtcttatg gatgcaattg tcacaggtga atggacaaca 180
ggtaatgcaa attcctcagt accagcatgt acaagaagga gaggacttca ccacgtactg 240
caattcctca actactttaa gcaatataca gtggtataag caaaggcctg gtggacatcc 300
cgtttttttg atacagttag tgaagagtgg agaagtgaag aagcagaaaa gactgacatt 360
tcagtttgga gaagcaaaaa agaacagctc cctgcacatc acagccaccc agactacaga 420
tgtaggaacc tacttctgtg cagggccgga caacttcaac aaattttact ttggatctgg 480
gaccaaactc aatgtaaaac ca 502
<210> 1070
<211> 417
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 42 - бета нативный
<400> 1070
gattcctgct gtgatcctgc catggatacc tggctcgtat gctgggcaat ttttagtctc 60
ttgaaagcag gactcacaga acctgaagtc acccagactc ccagccatca ggtcacacag 120
atgggacagg aagtgatctt gcgctgtgtc cccatctcta atcacttata cttctattgg 180
tacagacaaa tcttggggca gaaagtcgag tttctggttt ccttttataa taatgaaatc 240
tcagagaagt ctgaaatatt cgatgatcaa ttctcagttg aaaggcctga tggatcaaat 300
ttcactctga agatccggtc cacaaagctg gaggactcag ccatgtactt ctgtgccaaa 360
acatctcgaa gctcctacga gcagtacttc gggccgggca ccaggctcac ggtcaca 417
<210> 1071
<211> 508
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 43 - альфа нативный
<400> 1071
gaaagcagat tctttttatg atttttaaag tagaaatatc cattccaggt gcatttttta 60
agggtttaaa atttgaatcc tcagtgaacc agggcagaga agaatgatga aatccttgag 120
agttttacta gtgatcctgt ggcttcagtt gagctgggtt tggagccaac agaaggaggt 180
ggagcagaat tctggacccc tcagtgttcc agagggagcc attgcctctc tcaactgcac 240
ttacagtgac cgaggttccc agtccttctt ctggtacaga caatattctg ggaaaagccc 300
tgagttgata atgtccatat actccaatgg tgacaaagaa gatggaaggt ttacagcaca 360
gctcaataaa gccagccagt atgtttctct gctcatcaga gactcccagc ccagtgattc 420
agccacctac ctctgtgccg tgaacgttcc cacctcagga acctacaaat acatctttgg 480
aacaggcacc aggctgaagg ttttagca 508
<210> 1072
<211> 420
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 43 - бета нативный
<400> 1072
gctcattcct gctgtgatcc tgccatggat acctggctcg tatgctgggc aatttttagt 60
ctcttgaaag caggactcac agaacctgaa gtcacccaga ctcccagcca tcaggtcaca 120
cagatgggac aggaagtgat cttgcgctgt gtccccatct ctaatcactt atacttctat 180
tggtacagac aaatcttggg gcagaaagtc gagtttctgg tttcctttta taataatgaa 240
atctcagaga agtctgaaat attcgatgat caattctcag ttgaaaggcc tgatggatca 300
aatttcactc tgaagatccg gtccacaaag ctggaggact cagccatgta cttctgtgcc 360
agcacccccc gggacagcta cgagcagtac ttcgggccgg gcaccaggct cacggtcaca 420
<210> 1073
<211> 482
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 44 - альфа нативный
<400> 1073
ggagtttcct tccctcaccc acatgaagtg tctaccttct gcagactcca atggctcagg 60
aactgggaat gcagtgccag gctcgtggta tcctgcagca gatgtgggga gttttccttc 120
tttatgtttc catgaagatg ggaggcacta caggacaaaa cattgaccag cccactgaga 180
tgacagctac ggaaggtgcc attgtccaga tcaactgcac gtaccagaca tctgggttca 240
acgggctgtt ctggtaccag caacatgctg gcgaagcacc cacatttctg tcttacaatg 300
ttctggatgg tttggaggag aaaggtcgtt tttcttcatt ccttagtcgg tctaaagggt 360
acagttacct ccttttgaag gagctccaga tgaaagactc tgcctcttac ctctgtgccc 420
aatattctgg gggttaccag aaagttacct ttggaactgg aacaaagctc caagtcatcc 480
ca 482
<210> 1074
<211> 435
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 44 - бета нативный
<400> 1074
gagaatgctt actacagaga caccagcccc aagctaggag atcctgccat gggcttcagg 60
ctcctctgct gtgtggcctt ttgtctcctg ggagcaggcc cagtggattc tggagtcaca 120
caaaccccaa agcacctgat cacagcaact ggacagcgag tgacgctgag atgctcccct 180
aggtctggag acctctctgt gtactggtac caacagagcc tggaccaggg cctccagttc 240
ctcattcagt attataatgg agaagagaga gcaaaaggaa acattcttga acgattctcc 300
gcacaacagt tccctgactt gcactctgaa ctaaacctga gctctctgga gctgggggac 360
tcagctttgt atttctgtgc cagcagcggg accccagata cgcagtattt tggcccaggc 420
acccggctga cagtg 435
<210> 1075
<211> 508
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 45 - альфа нативный
<400> 1075
gctttccaaa atgcacacat ctactttcca aaatagaccg caactatttc tattgatctg 60
gaagaagctt gtaccaggca acccatttag gagaagttgg atgaagaggg agagggagat 120
gctactcatc acatcaatgt tggtcttatg gatgcaattg tcacaggtga atggacaaca 180
ggtaatgcaa attcctcagt accagcatgt acaagaagga gaggacttca ccacgtactg 240
caattcctca actactttaa gcaatataca gtggtataag caaaggcctg gtggacatcc 300
cgtttttttg atacagttag tgaagagtgg agaagtgaag aagcagaaaa gactgacatt 360
tcagtttgga gaagcaaaaa agaacagctc cctgcacatc acagccaccc agactacaga 420
tgtaggaacc tacttctgtg caggttcctc tggggctggg agttaccaac tcactttcgg 480
gaaggggacc aaactctcgg tcatacca 508
<210> 1076
<211> 450
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 45 - бета нативный
<400> 1076
gacagtgatc ctgatctggt aaagctccca tcctgccctg accctgccat gggcaccagg 60
ctcctcttct gggtggcctt ctgtctcctg ggggcatatc acacaggagc tggagtctcc 120
cagtccccca gtaacaaggt cacagagaag ggaaaggatg tagagctcag gtgtgatcca 180
atttcaggtc atactgccct ttactggtac cgacagaggc tggggcaggg cctggagttt 240
ttaatttact tccaaggcaa cagtgcacca gacaaatcag ggctgcccag tgatcgcttc 300
tctgcagaga ggactgggga atccgtctcc actctgacga tccagcgcac acagcaggag 360
gactcggccg tgtatctctg tgccagcagc ctttacttgg ggaccaccgg ggagctgttt 420
tttggagaag gctctaggct gaccgtactg 450
<210> 1077
<211> 524
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 46 - альфа нативный
<400> 1077
ggaaactttt caaagccagg gacttgtcca atccaacctc ctcacagttc ctagctcctg 60
aggctcagcg cccttggctt ctgtccgccc agctcaaggt cctgcagcat tgccactgct 120
cagccatgct cctgctgctc gtcccagcgt tccaggtgat ttttaccctg ggaggaacca 180
gagcccagtc tgtgacccag cttgacagcc aagtccctgt ctttgaagaa gcccctgtgg 240
agctgaggtg caactactca tcgtctgttt cagtgtatct cttctggtat gtgcaatacc 300
ccaaccaagg actccagctt ctcctgaagt atttatcagg atccaccctg gttaaaggca 360
tcaacggttt tgaggctgaa tttaacaaga gtcaaacttc cttccacttg aggaaaccct 420
cagtccatat aagcgacacg gctgagtact tctgtgctgt gagcccttcc tcaggaacct 480
acaaatacat ctttggaaca ggcaccaggc tgaaggtttt agca 524
<210> 1078
<211> 449
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 46 - бета нативный
<400> 1078
acagtgaccc tgatctggta aagctcccat cctgccctga ctctgtcatg ggcaccaggc 60
tcctctgctg ggcagccctg tgcctcctgg gggcagatca cacaggtgct ggagtctccc 120
agacccccag taacaaggtc acagagaagg gaaaatatgt agagctcagg tgtgatccaa 180
tttcaggtca tactgccctt tactggtacc gacaaagcct ggggcagggc ccagagtttc 240
taatttactt ccaaggcacg ggtgcggcag atgactcagg gctgcccaac gatcggttct 300
ttgcagtcag gcctgaggga tccgtctcta ctctgaagat ccagcgcaca gagcgggggg 360
actcagccgt gtatctctgt gccagcagct tatatctggg gggttcggag acccagtact 420
tcgggccagg cacgcggctc ctggtgctc 449
<210> 1079
<211> 509
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 47 - альфа нативный
<400> 1079
gaaaagcaga ttctttttat gatttttaaa gtagaaatat ccattctagg tgcatttttt 60
aagggtttaa aatttgaatc ctcagtgaac cagggcagag aagaatgatg aaatccttga 120
gagttttact agtgatcctg tggcttcagt tgagctgggt ttggagccaa cagaaggagg 180
tggagcagaa ttctggaccc ctcagtgttc cagagggagc cattgcctct ctcaactgca 240
cttacagtga ccgaggttcc cagtccttct tctggtacag acaatattct gggaaaagcc 300
ctgagttgat aatgttcata tactccaatg gtgacaaaga agatggaagg tttacagcac 360
agctcaataa agccagccag tatgtttctc tgctcatcag agactcccag cccagtgatt 420
cagccaccta cctctgtgcc gtgaacctgc tttcgggttc tgcaaggcaa ctgacctttg 480
gatctgggac acaattgact gttttacct 509
<210> 1080
<211> 420
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 47 - бета нативный
<400> 1080
gctcattcct gctgtgatcc tgccatggat acctggctcg tatgctgggc aatttttagt 60
ctcttgaaag caggactcac agaacctgaa gtcacccaga ctcccagcca tcaggtcaca 120
cagatgggac aggaagtgat cttgcgctgt gtccccatct ctaatcactt atacttctat 180
tggtacagac aaatcttggg gcagaaagtc gagtttctgg tttcctttta taataatgaa 240
atctcagaga agtctgaaat attcgatgat caattctcag ttgaaaggcc tgatggatca 300
aatttcactc tgaagatccg gtccacaaag ctggaggact cagccatgta cttctgtgcc 360
atgacagggc ggactaccta cgagcagtac ttcgggccgg gcaccaggct cacggtcaca 420
<210> 1081
<211> 507
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 48 - альфа нативный
<400> 1081
aggacagatt tcttttatga ttcctacagc agaaaaatga gaaacgtttg ttattatttt 60
tttttcgtgt ttaaggtttg aatcctcagt gaaccagggc agaaaagaat gatgaaatcc 120
ttgagagttt tactggtgat cctgtggctt cagttaagct gggtttggag ccaacagaag 180
gaggtggagc aggatcctgg accactcagt gttccagagg gagccattgt ttctctcaac 240
tgcacttaca gcaacagtgc ttttcaatac ttcatgtggt acagacagta ttccagaaaa 300
ggccctgagt tgctgatgta cacatactcc agtggtaaca aagaagatgg aaggtttaca 360
gcacaggtcg ataaatccag caagtatatc tccttgttca tcagagactc acagcccagt 420
gattcagcca cctacctctg tgcaatgcgg attcagggag cccagaagct ggtatttggc 480
caaggaacca ggctgactat caaccca 507
<210> 1082
<211> 420
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 48 - бета нативный
<400> 1082
gctcattcct gctgtgatcc tgccatggat acctggctcg tatgctgggc aatttttagt 60
ctcttgaaag caggactcac agaacctgaa gtcacccaga ctcccagcca tcaggtcaca 120
cagatgggac aggaagtgat cttgcgctgt gtccccatct ctaatcactt atacttctat 180
tggtacagac aaatcttggg gcagaaagtc gagtttctgg tttcctttta taataatgaa 240
atctcagaga agtctgaaat attcgatgat caattctcag ttgaaaggcc tgatggatca 300
aatttcactc tgaagatccg gtccacaaag ctggaggact cagccatgta cttctgtgcc 360
agcacaggca gggtctccta cgagcagtac ttcgggccgg gcaccaggct cacggtcaca 420
<210> 1083
<211> 508
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 49 - альфа нативный
<400> 1083
gaaagcagat tctttttatg atttttaaag tagaaatatc cattctaggt gcatttttta 60
agggtttaaa atttgaatcc tcagtgaacc agggcagaga agaatgatga aatccttgag 120
agttttacta gtgatcctgt ggcttcagtt gagctgggtt tggagccaac agaaggaggt 180
ggagcagaat tctggacccc tcagtgttcc agagggagcc attgcctctc tcaactgcac 240
ttacagtgac cgaggttccc agtccttctt ctggtacaga caatattctg ggaaaagccc 300
tgagttgata atgttcatat actccaatgg tgacaaagaa gatggaaggt ttacagcaca 360
gctcaataaa gccagccagt atgtttctct gctcatcaga gactcccagc ccagtgattc 420
agccacctac ctctgtgccg tgaacatagg gacctcagga acctacaaat acatctttgg 480
aacaggcacc aggctgaagg ttttagca 508
<210> 1084
<211> 417
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 49 - бета нативный
<400> 1084
gattcctgct gtgatcctgc catggatacc tggctcgtat gctgggcaat ttttagtctc 60
ttgaaagcag gactcacaga acctgaagtc acccagactc ccagccatca ggtcacacag 120
atgggacagg aagtgatctt gcgctgtgtc cccatctcta atcacttata cttctattgg 180
tacagacaaa tcttggggca gaaagtcgag tttctggttt ccttttataa taatgaaatc 240
tcagagaagt ctgaaatatt cgatgatcaa ttctcagttg aaaggcctga tggatcaaat 300
ttcactctga agatccggtc cacaaagctg gaggactcag ccatgtactt ctgtgccagc 360
actccaaggt attcctacga gcagtacttc gggccgggca ccaggctcac ggtcaca 417
<210> 1085
<211> 419
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 50 - альфа нативный
<400> 1085
ggcaggtccc agtggggaga acaatgaaga catttgctgg attttcgttc ctgtttttgt 60
ggctgcagct ggactgtatg agtagaggag aggatgtgga gcagagtctt ttcctgagtg 120
tccgagaggg agacagctcc gttataaact gcacttacac agacagctcc tccacctact 180
tatactggta taagcaagaa cctggagcag gtctccagtt gctgacgtat attttttcaa 240
atatggacat gaaacaagac caaagactca ctgttctatt gaataaaaag gataaacatc 300
tgtctctgcg cattgcagac acccagactg gggactcagc tatctacttc tgtgcagaga 360
gtccccctgg aacctacaaa tacatctttg gaacaggcac caggctgaag gttttagca 419
<210> 1086
<211> 421
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 50 - бета нативный
<400> 1086
gagccccaag ctaggagatc ctgccatggg cttcaggctc ctctgctgtg tggccttttg 60
tctcctggga gcaggcccag tggattctgg agtcacacaa accccaaagc acctgatcac 120
agcaactgga cagcgagtga cgctgagatg ctcccctagg tctggagacc tctctgtgta 180
ctggtaccaa cagagcctgg accagggcct ccagttcctc attcactatt ataatggaga 240
agagagagca aaaggaaaca ttcttgaacg attctccgca caacagttcc ctgacttgca 300
ctctgaacta aacctgagct ctctggagct gggggactca gctttgtatt tctgtgccag 360
cagcgccacc agggacgcct atggctacac cttcggttcg gggaccaggt taaccgttgt 420
a 421
<210> 1087
<211> 511
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 51 - альфа нативный
<400> 1087
gaaagcagat tctttttatg atttttaaag tagaaatatc cattccaggt gcatttttta 60
agggtttaaa atttgaatcc tcagtgaacc agggcagaga agaatgatga aatccttgag 120
agttttacta gtgatcctgt ggcttcagtt gagctgggtt tggagccaac agaaggaggt 180
ggagcagaat tctggacccc tcagtgttcc agagggagcc attgcctctc tcaactgcac 240
ttacagtgac cgaggttccc agtccttctt ctggtacaga caatattctg ggaaaagccc 300
tgagttgata atgtccatat actccaatgg tgacaaagaa gatggaaggt ttacagcaca 360
gctcaataaa gccagccagt atgtttctct gctcatcaga gactcccagc ccagtgattc 420
agccacctac ctctgtgccg tgaacatgag aggtggaggt agcaactata aactgacatt 480
tggaaaagga actctcttaa ccgtgaatcc a 511
<210> 1088
<211> 417
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 51 - бета нативный
<400> 1088
gattcctgct gtgatcctgc catggatacc tggctcgtat gctgggcaat ttttagtctc 60
ttgaaagcag gactcacaga acctgaagtc acccagactc ccagccatca ggtcacacag 120
atgggacagg aagtgatctt gcgctgtgtc cccatctcta atcacttata cttctattgg 180
tacagacaaa tcttggggca gaaagtcgag tttctggttt ccttttataa taatgaaatc 240
tcagagaagt ctgaaatatt cgatgatcaa ttctcagttg aaaggcctga tggatcaaat 300
ttcactctga agatccggtc cacaaagctg gaggactcag ccatgtactt ctgtgccatc 360
gctagtaggg tatcctacga gcagtacttc gggccgggca ccaggctcac ggtcaca 417
<210> 1089
<211> 467
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 52 - альфа нативный
<400> 1089
gagcccagag aagtctgttc caggacgggc tctttcagga gcagctaaag tcaggggcca 60
tgtccaccat gtgatagaaa gacaagatgg tcctgaaatt ctccgtgtcc attctttgga 120
ttcagttggc atgggtgagc acccagctgc tggagcagag ccctcagttt ctaagcatcc 180
aagagggaga aaatctcact gtgtactgca actcctcaag tgttttttcc agcttacaat 240
ggtacagaca ggagcctggg gaaggtcctg tcctcctggt gacagtagtt acgggtggag 300
aagtgaagaa gctgaagaga ctaacctttc agtttggtga tgcaagaaag gacagttctc 360
tccacatcac tgcagcccag cctggtgata caggcctcta cctctgtgca ggagcgaggg 420
actacaagct cagctttgga gccggaacca cagtaactgt aagagca 467
<210> 1090
<211> 421
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 52 - бета нативный
<400> 1090
gagccccaag ctaggagatc ctgccatggg cttcaggctc ctctgctgtg tggccttttg 60
tctcctggga gcaggcccag tggattctgg agtcacacaa accccaaagc acctgatcac 120
agcaactgga cagcgagtga cgctgagatg ctcccctagg tctggagacc tctctgtgta 180
ctggtaccaa cagagcctgg accagggcct ccagttcctc attcagtatt ataatggaga 240
agagagagca aaaggaaaca ttcttgaacg attctccgca caacagttcc ctgacttgca 300
ctctgaacta aacctgagct ctctggagct gggggactca gctttgtatt tctgtgccag 360
cagcgtaggg gacttaaaca atgagcagtt cttcgggcca gggacacggc tcaccgtgct 420
a 421
<210> 1091
<211> 416
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 53- альфа нативный
<400> 1091
gcttagctgg agccatggcc tctgcaccca tctcgatgct tgcgatgctc ttcacattga 60
gtgggctgag agctcagtca gtggctcagc cggaagatca ggtcaacgtt gctgaaggga 120
atcctctgac tgtgaaatgc acctattcag tctctggaaa cccttatctt ttttggtatg 180
ttcaataccc caaccgaggc ctccagttcc ttctgaaata catcacaggg gataacctgg 240
ttaaaggcag ctatggcttt gaagctgaat ttaacaagag ccaaacctcc ttccacctga 300
agaaaccatc tgcccttgtg agcgactccg ctttgtactt ctgtgctgtg agagactttg 360
gctcaggaac ctacaaatac atctttggaa caggcaccag gctgaaggtt ttagca 416
<210> 1092
<211> 453
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 53 - бета нативный
<400> 1092
gacagtgaca ctgatctggt aaagccctca tcctgtcctg accctgccat gggcaccagt 60
ctcctatgct gggtggtcct gggtttccta gggacagatc acacaggtgc tggagtctcc 120
cagtctccca ggtacaaagt cacaaagagg ggacaggatg tagctctcag gtgtgatcca 180
atttcgggtc atgtatccct ttattggtac cgacaggccc tggggcaggg cccagagttt 240
ctgacttact tcaattatga agcccaacaa gacaaatcag ggctgcccaa tgatcggttc 300
tctgcagaga ggcctgaggg atccatctcc actctgacga tccagcgcac agagcagcgg 360
gactcggcca tgtatcgctg tgccagcagc ggctccggga ctagcggcta caatgagcag 420
ttcttcgggc cagggacacg gctcaccgtg cta 453
<210> 1093
<211> 440
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 54 - альфа нативный
<400> 1093
gaagtcaact tctgggagca gatctctgca gaataaaaat gaaaaagcat ctgacgacct 60
tcttggtgat tttgtggctt tatttttata gggggaatgg caaaaaccaa gtggagcaga 120
gtcctcagtc cctgatcatc ctggagggaa agaactgcac tcttcaatgc aattatacag 180
tgagcccctt cagcaactta aggtggtata agcaagatac tgggagaggt cctgtttccc 240
tgacaatcat gactttcagt gagaacacaa agtcgaacgg aagatataca gcaactctgg 300
atgcagacac aaagcaaagc tctctgcaca tcacagcctc ccagctcagc gattcagcct 360
cctacatctg tgtggtgagc cgggataact atggtcagaa ttttgtcttt ggtcccggaa 420
ccagattgtc cgtgctgccc 440
<210> 1094
<211> 417
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 54 - бета нативный
<400> 1094
gattcctgct gtgatcctgc catggatacc tggctcgtat gctgggcaat ttttagtctc 60
ttgaaagcag gactcacaga acctgaagtc acccagactc ccagccatca ggtcacacag 120
atgggacagg aagtgatctt gcgctgtgtc cccatctcta atcacttata cttctattgg 180
tacagacaaa tcttggggca gaaagtcgag tttctggttt ccttttataa taatgaaatc 240
tcagagaagt ctgaaatatt cgatgatcaa ttctcagttg aaaggcctga tggatcaaat 300
ttcactctga agatccggtc cacaaagctg gaggactcag ccatgtactt ctgtgccatt 360
acagaccgca ctaactatgg ctacaccttc ggttcgggga ccaggttaac cgttgta 417
<210> 1095
<211> 507
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 55 - альфа нативный
<400> 1095
gagatgcctc tgtggacagt ttaagaaacc acagtgcttt ggaggaaagg aagagatact 60
tgataatata gctctcttgg ctggagattg caggtcccag tggggagaac aatgaagaca 120
tttgctggat tttcgttcct gtttttgtgg ctgcagctgg actgtatgag tagaggagag 180
gatgtggagc agagtctttt cctgagtgtc cgagagggag acagctccgt tataaactgc 240
acttacacag acagctcctc cacctactta tactggtata agcaagaacc tggagcaggt 300
ctccagttgc tgacgtatat tttttcaaat atggacatga aacaagacca aagactcact 360
gttctattga ataaaaagga taaacatctg tctctgcgca ttgcagacac ccagactggg 420
gactcagcta tctacttctg tgcagtcccc tcgggtgcta caaacaagct catctttgga 480
actggcactc tgcttgctgt ccagcca 507
<210> 1096
<211> 66
<212> ДНК
<213> Искусственная последовательность
<220>
<220>
<223> TCR 50/ TCR 54 P2A
<400> 1096
ggcagcggcg ccaccaactt cagcctgctt aaacaggcag gcgacgtgga ggagaatcct 60
ggccca 66
<210> 1097
<211> 402
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 15 альфа, кодон-оптимизированный/ цистеин-модифицированный
<400> 1097
atgagcctga gcagcctgct gaaggtggtc accgcttctc tgtggctggg ccctggaatc 60
gcccagaaga tcacccagac ccagcccggc atgttcgtgc aggaaaaaga ggccgtcacc 120
ctggactgca cctacgacac cagcgaccag agctacggcc tgttctggta caagcagccc 180
agcagcggcg agatgatctt cctgatctac cagggcagct acgacgagca gaacgccacc 240
gagggccggt acagcctgaa cttccagaag gcccggaagt ccgccaacct ggtcatctct 300
gccagccagc tgggcgacag cgccatgtac ttttgcgcca tgaagcctgg cggctacaac 360
aagctgatct tcggagccgg caccagactg gccgtgcacc ct 402
<210> 1098
<211> 402
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 15 бета, кодон-оптимизированный/ цистеин-модифицированный
<400> 1098
atggacagct ggaccttttg ctgcgtgtcc ctgtgcatcc tggtggccaa gcacaccgat 60
gccggcgtga tccagagccc cagacacgaa gtgaccgaga tgggccagga agtgaccctg 120
cgctgcaagc ccatcagcgg ccacaacagc ctgttctggt acagacagac catgatgcgg 180
ggcctggaac tgctgatcta cttcaacaac aacgtgccca tcgacgacag cggcatgccc 240
gaggacagat tcagcgccaa gatgcccaac gccagcttca gcaccctgaa gatccagccc 300
agcgagccca gagacagcgc cgtgtacttt tgcgccagca gcctcgtggg cagaagccgg 360
accgagcagt actttggccc tggcaccaga ctgaccgtga cc 402
<210> 1099
<211> 393
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 16 альфа, кодон-оптимизированный/ цистеин-модифицированный
<400> 1099
atggaaaccc tgctgggcct gctgatcctg tggctgcagc tccagtgggt gtccagcaag 60
caggaagtga cccagatccc tgccgccctg tctgtgcctg agggcgagaa cctggtgctg 120
aactgcagct tcaccgacag cgccatctac aacctgcagt ggttccggca ggaccccggc 180
aagggactga caagcctgct gctgattcag agcagccaga gagagcagac cagcggcaga 240
ctgaacgcca gcctggacaa gtcctccggc agaagcaccc tgtatatcgc cgcctcccag 300
cctggcgata gcgccacata tctgtgtgcc gtgcggcctg ccaacaacaa cgacatgaga 360
ttcggagccg gcacccggct gaccgtgaag cct 393
<210> 1100
<211> 399
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 16 бета, кодон-оптимизированный/ цистеин-модифицированный
<400> 1100
atgggcatca gactgctgtg cagagtggcc ttctgcttcc tggccgtggg cctggtggac 60
gtgaaagtga cccagagcag cagatacctg gtcaagcgga ccggcgagaa ggtgttcctg 120
gaatgcgtgc aggacatgga ccacgagaat atgttctggt acagacagga ccccggcctg 180
ggcctgcggc tgatctactt cagctacgac gtgaagatga aggaaaaggg cgacatcccc 240
gagggctaca gcgtgtccag agagaagaaa gagcggttca gcctgatcct ggaaagcgcc 300
agcaccaacc agaccagcat gtacctgtgc gccagcagcc tgtggggcag aagcaaccag 360
cctcagcact tcggcgacgg caccagactg agcatcctc 399
<210> 1101
<211> 402
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 17 альфа, кодон-оптимизированный/ цистеин-модифицированный
<400> 1101
atgagcctga gcagcctgct gaaggtggtc accgcttctc tgtggctggg ccctggaatc 60
gcccagaaga tcacccagac ccagcccggc atgttcgtgc aggaaaaaga ggccgtcacc 120
ctggactgca cctacgacac cagcgacccc agctacggcc tgttctggta caagcagccc 180
agcagcggcg agatgatctt cctgatctac cagggcagct acgaccagca gaacgccacc 240
gagggccggt acagcctgaa cttccagaag gcccggaagt ccgccaacct ggtcatctct 300
gccagccagc tgggcgacag cgccatgtac ttttgcgcca tgagagaggg cagaggcgac 360
aagatcattt tcggcaaggg cacccggctg cacatcctgc cc 402
<210> 1102
<211> 399
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 17 бета, кодон-оптимизированный/ цистеин-модифицированный
<400> 1102
atgggcatca gactgctgtg cagagtggcc ttctgcttcc tggccgtggg cctggtggac 60
gtgaaagtga cccagagcag cagatacctg gtcaagcgga ccggcgagaa ggtgttcctg 120
gaatgcgtgc aggacatgga ccacgagaat atgttctggt acagacagga ccccggcctg 180
ggcctgcggc tgatctactt cagctacgac gtgaagatga aggaaaaggg cgacatcccc 240
gagggctaca gcgtgtccag agagaagaaa gagcggttca gcctgatcct ggaaagcgcc 300
agcaccaacc agaccagcat gtacctgtgc gccagcagcc tgtggggcag aagcaaccag 360
cctcagcact tcggcgacgg caccagactg agcatcctc 399
<210> 1103
<211> 402
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 18 альфа, кодон-оптимизированный/ цистеин-модифицированный
<400> 1103
atggaaaccc tgctgggcct gctgatcctg tggctgcagc tccagtgggt gtccagcaag 60
caggaagtga cccagatccc tgccgccctg tctgtgcctg agggcgagaa cctggtgctg 120
aactgcagct tcaccgacag cgccatctac aacctgcagt ggttccggca ggaccccggc 180
aagggactga caagcctgct gctgattcag agcagccaga gagagcagac cagcggcaga 240
ctgaacgcca gcctggacaa gtcctccggc agaagcaccc tgtatatcgc cgcctcccag 300
cctggcgata gcgccacata tctgtgcgcc gagggcaatg ccggcggaac cagctacggc 360
aagctgacct ttggccaggg caccatcctg accgtgcacc cc 402
<210> 1104
<211> 399
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 18 бета, кодон-оптимизированный/ цистеин-модифицированный
<400> 1104
atgggcatca gactgctgtg cagagtggcc ttctgcttcc tggccgtggg cctggtggac 60
gtgaaagtga cccagagcag cagatacctg gtcaagcgga ccggcgagaa ggtgttcctg 120
gaatgcgtgc aggacatgga ccacgagaat atgttctggt acagacagga ccccggcctg 180
ggcctgcggc tgatctactt cagctacgac gtgaagatga aggaaaaggg cgacatcccc 240
gagggctaca gcgtgtccag agagaagaaa gagcggttca gcctgatcct ggaaagcgcc 300
agcaccaacc agaccagcat gtacctgtgc gccagcagct tctggggcag aagcaacagc 360
cctctgcact tcggcaacgg caccagactg accgtgacc 399
<210> 1105
<211> 408
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 19 альфа, кодон-оптимизированный/ цистеин-модифицированный
<400> 1105
atgagcctga gcagcctgct gaaggtggtc accgcttctc tgtggctggg ccctggaatc 60
gcccagaaga tcacccagac ccagcccggc atgttcgtgc aggaaaaaga ggccgtcacc 120
ctggactgca cctacgacac cagcgaccag agctacggcc tgttctggta caagcagccc 180
agcagcggcg agatgatctt cctgatctac cagggcagct acgacgagca gaacgccacc 240
gagggccggt acagcctgaa cttccagaag gcccggaagt ccgccaacct ggtcatctct 300
gccagccagc tgggcgacag cgccatgtac ttttgcgcca tgagagagaa caccggcacc 360
gccagcaagc tgaccttcgg cacaggcacc agactgcagg tcacactg 408
<210> 1106
<211> 399
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 19 бета, кодон-оптимизированный/ цистеин-модифицированный
<400> 1106
atgggcatca gactgctgtg cagagtggcc ttctgcttcc tggccgtggg cctggtggac 60
gtgaaagtga cccagagcag cagatacctg gtcaagcgga ccggcgagaa ggtgttcctg 120
gaatgcgtgc aggacatgga ccacgagaat atgttctggt acagacagga ccccggcctg 180
ggcctgcggc tgatctactt cagctacgac gtgaagatga aggaaaaggg cgacatcccc 240
gagggctaca gcgtgtccag agagaagaaa gagcggttca gcctgatcct ggaaagcgcc 300
agcaccaacc agaccagcat gtacctgtgc gccagcagca gctggggcca gagcacaggc 360
gagcagtact ttggccctgg caccagactg accgtgacc 399
<210> 1107
<211> 405
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 20 альфа, кодон-оптимизированный/ цистеин-модифицированный
<400> 1107
atgaacatgc tgaccgccag cctgctgaga gccgtgatcg ccagcatctg cgtggtgtcc 60
agcatggccc agaaagtgac ccaggcccag accgagatca gcgtggtgga aaaagaagat 120
gtgaccctgg actgcgtgta cgagacacgg gacaccacct actacctgtt ctggtacaag 180
cagcctccca gcggcgagct ggtgttcctg atcagacgga acagcttcga cgagcagaac 240
gagattagcg gccggtacag ctggaacttc cagaagtcca ccagcagctt caacttcacc 300
atcaccgcct cccaggtggt ggacagcgcc gtgtactttt gtgccctgtg gaccggggcc 360
aacaacctgt tctttggcac cggaacccgg ctgaccgtga tcccc 405
<210> 1108
<211> 393
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 20 бета, кодон-оптимизированный/ цистеин-модифицированный
<400> 1108
atgctgctgc tcctgctcct cctgggacct ggatctggac tgggagccgt ggtgtcccag 60
caccccagca gagtgatctg caagagcggc accagcgtga agatcgagtg cagaagcctg 120
gacttccagg ccaccaccat gttctggtac agacagttcc ccaagcagag cctgatgctg 180
atggccacct ccaacgaggg cagcaaggcc acatacgagc agggcgtgga aaaggacaag 240
ttcctgatca accacgccag cctgaccctg agcaccctga cagtgacaag cgcccacccc 300
gaggacagca gcttctacat ctgtagcgcc agcagcctgg ccagaagaag ctacgagcag 360
tacttcggcc ctggcaccag actgaccgtg acc 393
<210> 1109
<211> 414
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 21 альфа, кодон-оптимизированный/ цистеин-модифицированный
<400> 1109
atgagcctga gcagcctgct gaaggtggtc accgcttctc tgtggctggg ccctggaatc 60
gcccagaaga tcacccagac ccagcccggc atgttcgtgc aggaaaaaga ggccgtcacc 120
ctggactgca cctacgacac cagcgaccag agctacggcc tgttctggta caagcagccc 180
agcagcggcg agatgatctt cctgatctac cagggcagct acgacgagca gaacgccacc 240
gagggccggt acagcctgaa cttccagaag gcccggaagt ccgccaacct ggtcatctct 300
gccagccagc tgggcgacag cgccatgtac ttttgcgcca tgcgcgaggg cagagtgacc 360
ggcggaggca acaagctgac ctttggcacc ggcacccagc tgaaagtgga actg 414
<210> 1110
<211> 441
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 21 бета, кодон-оптимизированный/ цистеин-модифицированный
<400> 1110
atgcacagac ctagaaggcc cctgcatccc gtggcccctg ccatgtctat cggcctgctg 60
tgctgcgtgg cattctccct gctgtgggcc tctcctgtga atgccggcgt gacccagacc 120
cctaagttcc aggtgctgaa aaccggccag agcatgaccc tgcagtgcgc ccaggacatg 180
aaccacaaca gcatgtactg gtacagacag gaccccggca tgggcctgcg gctgatctac 240
tatagcgcca gcgagggcac caccgacaag ggcgaggtgc ccaacggcta caacgtgtcc 300
cggctgaaca agagagagtt cagcctgaga ctggaaagcg ccgctcccag ccagaccagc 360
gtgtacttct gtgccagcag accctggggc aaccagaaca ccgaggcatt ctttgggcag 420
ggcacccggc tgacagtggt c 441
<210> 1111
<211> 438
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 22 альфа, кодон-оптимизированный/ цистеин-модифицированный
<400> 1111
atggcccagg aactgggcat gcagtgtcag gccagaggca tcctgcagca gatgtggggc 60
gtgttcctgc tgtacgtgtc catgaagatg ggcggcacca ccggccagaa catcgaccag 120
cctaccgaga tgaccgccac cgagggcgct atcgtgcaga tcaactgcac ctaccagacc 180
agcggcttca acggcctgtt ctggtatcag cagcacgccg gcgaggcccc taccttcctg 240
agctacaatg tgctggacgg cctggaagag aagggccggt tcagcagctt cctgtccaga 300
agcaagggct acagctacct gctgctgaaa gaactgcaga tgaaggacag cgcctcctac 360
ctgtgtgccg tgcgggccag aatggacagc aactaccagc tgatctgggg agccggcacc 420
aagctgatca tcaagccc 438
<210> 1112
<211> 393
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 22 бета, кодон-оптимизированный/ цистеин-модифицированный
<400> 1112
atgggacctg gactgctgtg ctgggtgctg ctgtgtctgc tgggagccgg acctgtggat 60
gccggcgtga cacagagccc cacccacctg atcaagacca gaggccagca cgtgaccctg 120
cggtgtagcc ctatcagcgg ccacaagagc gtgtcctggt atcagcaggt cctgggccag 180
ggccctcagt tcatcttcca gtactacgag aaagaggaac gcggcagagg caacttcccc 240
gacagattca gcgccagaca gttccccaac tacagcagcg agctgaacgt gaacgccctg 300
ctgctgggcg atagcgccct gtacctgtgt gccagcagcc ggaccgagaa ctacggctac 360
accttcggca gcggcaccag actgacagtg gtc 393
<210> 1113
<211> 408
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 23 альфа, кодон-оптимизированный/ цистеин-модифицированный
<400> 1113
atgagcctga gcagcctgct gaaggtggtc accgcttctc tgtggctggg ccctggaatc 60
gcccagaaga tcacccagac ccagcccggc atgttcgtgc aggaaaaaga ggccgtcacc 120
ctggactgca cctacgacac cagcgaccag agctacggcc tgttctggta caagcagccc 180
agcagcggcg agatgatctt cctgatctac cagggcagct acgacgagca gaacgccacc 240
gagggccggt acagcctgaa cttccagaag gcccggaagt ccgccaacct ggtcatctct 300
gccagccagc tgggcgacag cgccatgtac ttttgcgcca tgagcccacc tggcggcagc 360
gccagacagc tgacatttgg ctctggcacc cagctgaccg tgctgccc 408
<210> 1114
<211> 399
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 23 бета, кодон-оптимизированный/ цистеин-модифицированный
<400> 1114
atgggcatca gactgctgtg cagagtggcc ttctgcttcc tggccgtggg cctggtggac 60
gtgaaagtga cccagagcag cagatacctg gtcaagcgga ccggcgagaa ggtgttcctg 120
gaatgcgtgc aggacatgga ccacgagaat atgttctggt acagacagga ccccggcctg 180
ggcctgcggc tgatctactt cagctacgac gtgaagatga aggaaaaggg cgacatcccc 240
gagggctaca gcgtgtccag agagaagaaa gagcggttca gcctgatcct ggaaagcgcc 300
agcaccaacc agaccagcat gtacctgtgc gccagcagcc cctggggcca gtctaatcag 360
cctcagcact tcggcgacgg cacccggctg tctattctc 399
<210> 1115
<211> 411
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 24 альфа, кодон-оптимизированный/ цистеин-модифицированный
<400> 1115
atgagcctga gcagcctgct gaaggtggtc accgcttctc tgtggctggg ccctggaatc 60
gcccagaaga tcacccagac ccagcccggc atgttcgtgc aggaaaaaga ggccgtcacc 120
ctggactgca cctacgacac cagcgaccag agctacggcc tgttctggta caagcagccc 180
agcagcggcg agatgatctt cctgatctac cagggcagct acgacgagca gaacgccacc 240
gagggccggt acagcctgaa cttccagaag gcccggaagt ccgccaacct ggtcatctct 300
gccagccagc tgggcgacag cgccatgtac ttttgcgcca tgagagaggg cagaggcgac 360
agctggggca agctgcagtt tggagccggc acccaggtgg tcgtgacccc t 411
<210> 1116
<211> 399
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 24 бета, кодон-оптимизированный/ цистеин-модифицированный
<400> 1116
atgggcatca gactgctgtg cagagtggcc ttctgcttcc tggccgtggg cctggtggac 60
gtgaaagtga cccagagcag cagatacctg gtcaagcgga ccggcgagaa ggtgttcctg 120
gaatgcgtgc aggacatgga ccacgagaat atgttctggt acagacagga ccccggcctg 180
ggcctgcggc tgatctactt cagctacgac gtgaagatga aggaaaaggg cgacatcccc 240
gagggctaca gcgtgtccag agagaagaaa gagcggttca gcctgatcct ggaaagcgcc 300
agcaccaacc agaccagcat gtacctgtgc gccagcagcc ctttcggcag aggcagctac 360
gagcagtact tcggccctgg caccagactg accgtgacc 399
<210> 1117
<211> 414
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 25 альфа, кодон-оптимизированный/ цистеин-модифицированный
<400> 1117
atgagcctga gcagcctgct gaaggtggtc accgcttctc tgtggctggg ccctggaatc 60
gcccagaaga tcacccagac ccagcccggc atgttcgtgc aggaaaaaga ggccgtcacc 120
ctggactgca cctacgacac cagcgaccag agctacggcc tgttctggta caagcagccc 180
agcagcggcg agatgatctt cctgatctac cagggcagct acgacgagca gaacgccacc 240
gagggccggt acagcctgaa cttccagaag gcccggaagt ccgccaacct ggtcatctct 300
gccagccagc tgggcgacag cgccatgtac ttttgcgcca tgagagaggg cagcctgacc 360
ggcggaggca acaagctgac ctttggcacc ggcacccagc tgaaagtgga actg 414
<210> 1118
<211> 399
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 25 бета, кодон-оптимизированный/ цистеин-модифицированный
<400> 1118
atgggcatca gactgctgtg cagagtggcc ttctgcttcc tggccgtggg cctggtggac 60
gtgaaagtga cccagagcag cagatacctg gtcaagcgga ccggcgagaa ggtgttcctg 120
gaatgcgtgc aggacatgga ccacgagaat atgttctggt acagacagga ccccggcctg 180
ggcctgcggc tgatctactt cagctacgac gtgaagatga aggaaaaggg cgacatcccc 240
gagggctaca gcgtgtccag agagaagaaa gagcggttca gcctgatcct ggaaagcgcc 300
agcaccaacc agaccagcat gtacctgtgc gccagcagcc tgtggggcca gagcaatcag 360
cctcagcact tcggcgacgg cacccggctg tctattctc 399
<210> 1119
<211> 402
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 26 альфа, кодон-оптимизированный/ цистеин-модифицированный
<400> 1119
atgagcctga gcagcctgct gaaggtggtc accgcttctc tgtggctggg ccctggaatc 60
gcccagaaga tcacccagac ccagcccggc atgttcgtgc aggaaaaaga ggccgtcacc 120
ctggactgca cctacgacac cagcgaccag agctacggcc tgttctggta caagcagccc 180
agcagcggcg agatgatctt cctgatctac cagggcagct acgacgagca gaacgccacc 240
gagggccggt acagcctgaa cttccagaag gcccggaagt ccgccaacct ggtcatctct 300
gccagccagc tgggcgacag cgccatgtac ttttgcgcca tgcgggacgc ccggaacaac 360
gacatgagat tcggagccgg cacccggctg accgtgaagc ct 402
<210> 1120
<211> 399
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 26 бета, кодон-оптимизированный/ цистеин-модифицированный
<400> 1120
atgtctctgg gactgctgtg ctgcggagcc ttctctctgc tgtgggccgg acctgtgaat 60
gccggcgtga cccagacccc taagttccgg gtgctgaaaa ccggccagag catgaccctg 120
ctgtgcgccc aggacatgaa ccacgagtac atgtattggt acagacagga ccccggcatg 180
ggcctgcggc tgatccacta ttctgtgggc gagggcacca ccgccaaggg cgaagtgcct 240
gacggctaca acgtgtcccg gctgaagaag cagaacttcc tgctgggcct ggaaagcgcc 300
gctcctagcc agaccagcgt gtacttctgc gccagcagct actacgccag cggccggaac 360
tacgagcagt acttcggccc tggcacccgg ctgacagtg 399
<210> 1121
<211> 393
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 27 альфа, кодон-оптимизированный/ цистеин-модифицированный
<400> 1121
atgaagctgg tcaccagcat caccgtgctg ctgagcctgg gaatcatggg cgacgccaag 60
accacccagc ccaacagcat ggaaagcaac gaagaggaac ccgtgcatct gccctgcaac 120
cacagcacca tcagcggcac cgactacatc cactggtaca gacagctgcc cagccagggc 180
cccgagtatg tgatccacgg cctgaccagc aacgtgaaca accggatggc ctccctggcc 240
attgccgagg acagaaagag cagcaccctg atcctgcacc gggccacact gagagatgcc 300
gccgtgtact actgcatcct gcgggtgcca cctcagagcg gcggctacca gaaagtgacc 360
ttcggcaccg gcaccaagct gcaggtcatc ccc 393
<210> 1122
<211> 441
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 27 бета, кодон-оптимизированный/ цистеин-модифицированный
<400> 1122
atgcacagac ctagaaggcc cctgcatccc gtggcccctg ccatgtctat cggcctgctg 60
tgctgcgtgg cattctccct gctgtgggcc tctcctgtga atgccggcgt gacccagacc 120
cctaagttcc aggtgctgaa aaccggccag agcatgaccc tgcagtgcgc ccaggacatg 180
aaccacaaca gcatgtactg gtacagacag gaccccggca tgggcctgcg gctgatctac 240
tatagcgcca gcgagggcac caccgacaag ggcgaggtgc ccaacggcta caacgtgtcc 300
cggctgaaca agagagagtt cagcctgaga ctggaaagcg ccgctcccag ccagaccagc 360
gtgtactttt gcgccagcag cgagttcggc agcctgaacg agaagctgtt cttcggcagc 420
ggcacccagc tgagcgtgct c 441
<210> 1123
<211> 402
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 28 альфа, кодон-оптимизированный/ цистеин-модифицированный
<400> 1123
atgatgatca gcctgcgggt gctgctggtc atcctgtggc tgcagctgag ctgggtctgg 60
tcccagcgga aagaggtgga acaggaccct ggccccttca acgtgccaga gggcgctacc 120
gtggccttca actgcaccta cagcaacagc gccagccaga gcttcttctg gtacagacag 180
gactgccgga aagaacccaa gctgctgatg agcgtgtaca gcagcggcaa cgaggacggc 240
agattcaccg cccagctgaa ccgggccagc cagtacatct ccctgctgat ccgggacagc 300
aagctgagcg acagcgccac ctatctgtgt gtcgtgcggg gaggcggcac cagctacggc 360
aagctgacat ttggccaggg caccatcctg accgtgcacc cc 402
<210> 1124
<211> 402
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 28 бета, кодон-оптимизированный/ цистеин-модифицированный
<400> 1124
atgggcatca gactgctgtg cagagtggcc ttctgcttcc tggccgtggg cctggtggac 60
gtgaaagtga cccagagcag cagatacctg gtcaagcgga ccggcgagaa ggtgttcctg 120
gaatgcgtgc aggacatgga ccacgagaat atgttctggt acagacagga ccccggcctg 180
ggcctgcggc tgatctactt cagctacgac gtgaagatga aggaaaaggg cgacatcccc 240
gagggctaca gcgtgtccag agagaagaaa gagcggttca gcctgatcct ggaaagcgcc 300
agcaccaacc agaccagcat gtacctgtgc gccagcagcc tgtggggcag aagcagcggc 360
aacaccatct acttcggcga gggcagctgg ctgaccgtgg tc 402
<210> 1125
<211> 408
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 29 альфа, кодон-оптимизированный/ цистеин-модифицированный
<400> 1125
atgagcctga gcagcctgct gaaggtggtc accgcttctc tgtggctggg ccctggaatc 60
gcccagaaga tcacccagac ccagcccggc atgttcgtgc aggaaaaaga ggccgtcacc 120
ctggactgca cctacgacac cagcgaccag agctacggcc tgttctggta caagcagccc 180
agcagcggcg agatgatctt cctgatctac cagggcagct acgacgagca gaacgccacc 240
gagggccggt acagcctgaa cttccagaag gcccggaagt ccgccaacct ggtcatctct 300
gccagccagc tgggcgacag cgccatgtac ttttgcgcca tgcggctgaa caccggcacc 360
gccagcaagc tgaccttcgg cacaggcacc agactgcagg tcacactg 408
<210> 1126
<211> 399
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 29 бета, кодон-оптимизированный/ цистеин-модифицированный
<400> 1126
atgagcaatc aggtgctgtg ctgcgtggtc ctgtgcctgc tgggagccaa taccgtggat 60
ggcggcatca cccagagccc caagtacctg ttccggaaag agggccagaa cgtcaccctg 120
agctgcgagc agaacctgaa ccacgacgcc atgtactggt acagacagga ccccggccag 180
ggcctgcggc tgatctacta cagccagatc gtgaacgact tccagaaggg cgacattgcc 240
gagggctaca gcgtgtccag agagaagaaa gagtccttcc cactgaccgt gaccagcgcc 300
cagaagaacc ccaccgcctt ctacctgtgt gccagctctc cttggggcag agccaccaac 360
gagcagttct tcggccctgg caccaggctg acagtgctg 399
<210> 1127
<211> 393
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 30 альфа, кодон-оптимизированный/ цистеин-модифицированный
<400> 1127
atgcggctgg tggccagagt gaccgtgttc ctgaccttcg gcaccatcat cgacgccaag 60
accacccagc ctcccagcat ggattgtgcc gagggcagag ccgccaacct gccctgcaat 120
cacagcacca tcagcggcaa cgagtacgtg tactggtaca gacagatcca cagccagggc 180
cctcagtaca tcatccacgg cctgaagaac aacgagacaa acgagatggc cagcctgatc 240
atcaccgagg acagaaagag cagcaccctg atcctgcccc acgccaccct gagagacacc 300
gccgtgtact actgtatcgt gcggggcaca agcgtgctgc agggcaacga gaagctgaca 360
tttggcaccg gcacccggct gaccatcatc ccc 393
<210> 1128
<211> 402
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 30 бета, кодон-оптимизированный/ цистеин-модифицированный
<400> 1128
atggatacct ggctcgtgtg ctgggccatc ttcagcctgc tgaaggccgg actgaccgag 60
cccgaagtga cccagacacc cagccaccag gtcacacaga tgggccagga agtgatcctg 120
cgctgcgtgc ccatcagcaa ccacctgtac ttctactggt acagacagat cctgggccag 180
aaggtggaat tcctcgtcag cttctacaac aacgagatca gcgagaagtc cgagattttc 240
gacgaccagt tcagcgtgga acggcccgac ggcagcaact tcaccctgaa gatcagaagc 300
accaagctcg aggacagcgc catgtacttt tgcgccagca gcagacagcc cagcagcggc 360
aacaccatct acttcggcga gggcagctgg ctgaccgtgg tc 402
<210> 1129
<211> 414
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 31 альфа, кодон-оптимизированный/ цистеин-модифицированный
<400> 1129
atggccatgc tgctgggagc ctctgtgctg atcctgtggc tccagcccga ctgggtcaac 60
agccagcaga agaacgacga ccagcaggtc aaacagaaca gccccagcct gagcgtgcag 120
gaaggccgga tcagcatcct gaactgcgac tacaccaaca gcatgttcga ctactttctg 180
tggtacaaga agtaccccgc cgagggcccc accttcctga tctccatcag cagcatcaag 240
gacaagaacg aggacggccg gttcaccgtg tttctgaaca agagcgccaa gcacctgagc 300
ctgcacatcg tgcctagcca gcctggcgac agcgccgtgt acttttgtgc cgccagcgag 360
cggggcacct acaagtacat cttcggcacc ggcacccggc tgaaggtgct ggct 414
<210> 1130
<211> 402
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 31 бета, кодон-оптимизированный/ цистеин-модифицированный
<400> 1130
atgggcacca gactgctgtt ctgggtggcc ttctgcctgc tgggagccga tcatacaggt 60
gccggcgtgt cccagagccc cagcaacaaa gtgaccgaga agggcaagga cgtggaactg 120
agatgcgacc ccatcagcgg ccacaccgcc ctgtactggt acagacagtc tctgggccag 180
ggcctggaat tcctgatcta ttttcagggc aacagcgccc ctgacaagag cggcctgccc 240
agcgatagat tcagcgccga gagaacaggc ggcagcgtgt ccaccctgac catccagaga 300
acccagcagg aagatagcgc cgtctacctg tgcgccagca gcagattcct gggcagcacc 360
gacacccagt acttcggccc aggcaccagg ctgacagtgc tg 402
<210> 1131
<211> 381
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 32 альфа, кодон-оптимизированный/ цистеин-модифицированный
<400> 1131
atggtgctga agttcagcgt gtccatcctg tggatccagc tggcctgggt gtccacccag 60
ctgctggaac agagccctca gttcctgagc atccaggaag gcgagaacct gaccgtgtac 120
tgcaacagca gcagcgtgtt cagcagcctg cagtggtaca gacaggaacc cggcgaggga 180
cctgtgctgc tggtcacagt cgtgacaggc ggcgaagtga agaagctgaa gcggctgacc 240
ttccagttcg gcgacgccag aaaggacagc tccctgcaca ttacagccgc ccagcctggc 300
gataccggcc tgtatctgtg tgccggcgga agcaactaca agctgacctt tggcaagggc 360
accctgctga ccgtgaaccc c 381
<210> 1132
<211> 399
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 32 бета, кодон-оптимизированный/ цистеин-модифицированный
<400> 1132
atgggcttca gactgctgtg ctgcgtggcc ttctgtctgc tgggagccgg acctgtggat 60
agcggcgtga cccagacccc taagcacctg atcaccgcca ccggccagcg cgtgacactg 120
agatgtagcc ctagaagcgg cgacctgagc gtgtactggt atcagcagag cctggaccag 180
ggcctgcagt tcctgatcca gtactacaac ggcgaggaac gggccaaggg caacatcctg 240
gaacggttca gcgcccagca gttccccgat ctgcacagcg agctgaacct gagcagcctg 300
gaactgggcg acagcgccct gtacttctgt gcctcttctg tgggcggcga ccacagcgac 360
gagcagtttt ttggccctgg cacccggctg accgtgctg 399
<210> 1133
<211> 405
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 33 альфа, кодон-оптимизированный/ цистеин-модифицированный
<400> 1133
atgatgaagt ccctgcgggt gctgctggtc atcctgtggc tgcagctgag ctgggtctgg 60
tcccagcaga aagaggtgga acagaacagc ggcccactga gcgtgccaga gggcgctatc 120
gccagcctga actgcaccta cagcgaccgg ggcagccaga gcttcttctg gtacagacag 180
tacagcggca agagccccga gctgatcatg ttcatctaca gcaacggcga caaagaggac 240
ggccggttca ccgcccagct gaacaaggcc agccagtacg tgtccctgct gatcagagac 300
agccagccca gcgacagcgc cacctacctg tgtgccgtga atgcccacca caccggcggc 360
ttcaagacca tctttggagc cggcacccgg ctgttcgtga aggcc 405
<210> 1134
<211> 396
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 33 бета, кодон-оптимизированный/ цистеин-модифицированный
<400> 1134
atggatacct ggctcgtgtg ctgggccatc ttcagcctgc tgaaggccgg actgaccgag 60
cccgaagtga cccagacacc cagccaccag gtcacacaga tgggccagga agtgatcctg 120
cgctgcgtgc ccatcagcaa ccacctgtac ttctactggt acagacagat cctgggccag 180
aaggtggaat tcctcgtcag cttctacaac aacgagatca gcgagaagtc cgagattttc 240
gacgaccagt tcagcgtgga acggcccgac ggcagcaact tcaccctgaa gatcagaagc 300
accaagctcg aggacagcgc catgtacttt tgcgccagca cccctcggga cacctacgag 360
cagtacttcg gccctggcac cagactgacc gtgacc 396
<210> 1135
<211> 381
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 34 альфа, кодон-оптимизированный/ цистеин-модифицированный
<400> 1135
atgaagaaac tgctggccat gatcctgtgg ctgcagctgg acagactgag cggcgagctg 60
aaggtggaac agaaccctct gttcctgagc atgcaggaag gcaagaacta caccatctac 120
tgcaactaca gcaccaccag cgaccggctg tactggtaca gacaggaccc cggcaagagc 180
ctggaaagcc tgttcgtgct gctgagcaac ggggccgtga agcaggaagg acggctgatg 240
gccagcctgg acaccaaggc cagactgtcc accctgcaca tcacagccgc cgtgcacgac 300
ctgagcgcca cctacttttg tgccgtgtcc ggcacctaca agtacatctt cggcaccggc 360
accagactga aggtgctggc c 381
<210> 1136
<211> 396
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 34 бета, кодон-оптимизированный/ цистеин-модифицированный
<400> 1136
atgggacctg gactgctgtg ttgggccctg ctgtgtctgc tgggagccgg atctgtggaa 60
accggcgtga cccagagccc cacccacctg atcaagacca gaggccagca ggtcacactg 120
cggtgtagca gccagagcgg ccacaacacc gtgtcctggt atcagcaggc cctgggccag 180
ggaccccagt tcatcttcca gtactaccgc gaggaagaga acggcagagg caacttccca 240
ccccggttca gcggcctgca gttccccaat tacagcagcg agctgaacgt gaacgccctg 300
gaactggacg acagcgccct gtacctgtgc gcctctagca gctacgccgg cagctacgag 360
cagtacttcg gccctggcac cagactgacc gtgacc 396
<210> 1137
<211> 387
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 35 альфа, кодон-оптимизированный/ цистеин-модифицированный
<400> 1137
atggctctgc agtctacact gggagctgtg tggctgggcc tgctgctgaa ttctctgtgg 60
aaggtggccg agagcaagga ccaggtgttc cagccttcta cagtggcctc ttctgagggc 120
gccgtggtgg aaatcttctg caaccacagc gtgtccaacg cctacaactt cttctggtat 180
ctgcacttcc ccggctgcgc ccctagactg cttgtgaagg gaagcaagcc tagccagcag 240
ggcagataca acatgaccta cgagcggttc agcagctccc tgctgattct gcaagtgcgg 300
gaagccgatg ccgccgtgta ttattgtgct gtggccggcg actacaagct gagctttgga 360
gccggcacca cagtgacagt gcgggct 387
<210> 1138
<211> 402
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 35 бета, кодон-оптимизированный/ цистеин-модифицированный
<400> 1138
atgggcttta gactgctgtg ctgcgtggcc ttctgtctgc ttggagctgg ccctgtggat 60
agcggcgtta cccagacacc taagcacctg atcacagcca caggccagcg cgtgaccctg 120
agatgttctc ctagaagcgg cgacctgagc gtgtactggt atcagcagtc tctggaccag 180
ggcctgcagt tcctgatcca gtactacaac ggcgaggaaa gagccaaggg caacatcctg 240
gaacggttca gcgcccagca gttcccagat ctgcacagcg agctgaacct gagcagcctg 300
gaactgggag atagcgccct gtacttctgt gccagcacca caagcggcga tagcagctac 360
aacgagcagt tcttcggccc tggcaccaga ctgacagttc tg 402
<210> 1139
<211> 399
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 36 альфа, кодон-оптимизированный/ цистеин-модифицированный
<400> 1139
atgatgatca gcctgcgggt gctgctggtc atcctgtggc tgcaactgag ctgggtctgg 60
tcccagcgga aagaggtgga acaggacccc ggacctttca atgtgcctga aggcgccacc 120
gtggccttca actgcaccta cagcaatagc gccagccaga gcttcttctg gtacagacag 180
gactgccgga aagaacccaa gctgctgatg agcgtgtaca gcagcggcaa cgaggacggc 240
agattcacag cccagctgaa cagagccagc cagtacatct ccctgctgat ccgggatagc 300
aagctgagcg atagcgccac ctacctgtgc gtggtcaaca gagacaacta cggccagaac 360
ttcgtgttcg gccctggcac aagactgagc gtgctgcct 399
<210> 1140
<211> 396
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 36 бета, кодон-оптимизированный/ цистеин-модифицированный
<400> 1140
atggatactt ggcttgtgtg ctgggccatc ttcagcctgc tgaaggccgg actgacagag 60
cccgaagtga cacagacacc cagccaccaa gtgacccaga tgggccaaga agtgatcctg 120
cgctgcgtgc ccatcagcaa ccacctgtac ttctactggt acagacagat cctgggccag 180
aaagtggaat tcctggtgtc cttctacaac aacgagatca gcgagaagtc cgagatcttc 240
gacgaccagt tcagcgtgga aagacccgac ggcagcaact tcaccctgaa gatcagaagc 300
accaagctcg aggacagcgc catgtacttt tgcgccatga ccggcagaag caactacgag 360
cagtacttcg gccctggcac cagactgaca gtgaca 396
<210> 1141
<211> 390
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 37 альфа, кодон-оптимизированный/ цистеин-модифицированный
<400> 1141
atgaagaaac tgctggccat gatcctgtgg ctgcagctgg atagactgag cggcgagctg 60
aaggtggaac agaaccctct gttcctgagc atgcaagagg gcaagaacta caccatctac 120
tgcaactaca gcaccaccag cgaccggctg tactggtaca gacaggatcc aggcaagagc 180
ctggaaagcc tgttcgtgct gctgagcaat ggcgccgtga aacaagaggg tagactgatg 240
gccagcctgg acaccaaggc cagactgagc acactgcaca ttacagccgc cgtgcacgat 300
ctgagcgcca cctatttttg tgccggctac tctggcgccg gaagctacca gctgacattt 360
ggcaagggca ccaagctgag cgtgatcccc 390
<210> 1142
<211> 399
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 37 бета, кодон-оптимизированный/ цистеин-модифицированный
<400> 1142
atgggaacaa gactgctgtg ttgggtcgtg ctgggcttcc tgggcacaga tcatacaggt 60
gccggtgtct ctcagagccc cagatacaag gtggccaagc gcggacagga tgtggccctg 120
agatgtgatc ctatcagcgg ccacgtgtcc ctgttctggt atcagcaggc tctcggacag 180
ggccccgagt tcctgaccta ctttcagaat gaggcccagc tggacaagag cggcctgcct 240
agcgatagat tcttcgccga aagacccgag ggcagcgtgt ccacactgaa gatccagaga 300
acccagcaag aggacagcgc cgtgtacctg tgtgcctcta gtctgctgct gggcgcctac 360
ggctacacat ttggctctgg caccagactg accgtggtc 399
<210> 1143
<211> 414
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 38 альфа, кодон-оптимизированный/ цистеин-модифицированный
<400> 1143
atggtcaaga tgcctggcgc tagacggcag agcatcatga agagaattct gggcgccctg 60
ctgggcctgc tgtctgccca agtttgttgt gtgcggggca tccaggtgga acagagccct 120
cctgatctga tcctgcaaga gggcgccaac agcaccctga gatgcaactt cagcgacagc 180
gtgaacaacc tgcagtggtt ccaccagaat ccttggggcc agctgatcaa cctgttctac 240
atccccagcg gcaccaagca gaacggcaga ctgtctgcta ccaccgtggc caccgagaga 300
tacagcctgc tgtacatcag cagcagccag accacagaca gcggcgtgta cttttgcgcc 360
gtgggcttca acgacatgag attcggagcc ggcaccagac tgaccgtgaa gcct 414
<210> 1144
<211> 396
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 38 бета, кодон-оптимизированный/ цистеин-модифицированный
<400> 1144
atgggacctg gactgctttg ttgggtgctg ctgtgtctgc tcggagccgg atctgtggaa 60
acaggcgtga cacagagccc cacacacctg atcaagacca gaggccagca agtgaccctg 120
agatgcagct ctcagagcgg ccacaatacc gtgtcctggt atcagcaggc cctcggacag 180
ggccctcagt tcatcttcca gtactacaga gaggaagaga acggcagagg caacttccca 240
cctagattca gcggcctgca gttccccaac tacagcagcg agctgaacgt gaacgccctg 300
gaactggatg acagcgccct gtacctgtgt gcctcttctc ttgtggctgg cggcgagaca 360
cagtactttg gccccggaac aagactgctg gtgctg 396
<210> 1145
<211> 357
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 39 альфа, кодон-оптимизированный/ цистеин-модифицированный
<400> 1145
atgcagctga catgggtttc cggccagcag ctgaatcaga gccctcagag catgttcatc 60
caagaaggcg aggacgtttc catgaactgc accagcagca gcatcttcaa cacctggctg 120
tggtacaagc aagagcctgg cgaaggaccc gtgctgctga tcgctcttta caaggccggc 180
gagctgacca gcaacggcag actgacagcc cagttcggca ttacccggaa ggacagcttc 240
ctgaacatca gcgccagcat tcccagcgac gtgggcatct atttctgcgc cggctacagc 300
agctccaacg actacaagct gagcttcgga gccggcacca ccgtgacagt tagagcc 357
<210> 1146
<211> 399
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 39 бета, кодон-оптимизированный/ цистеин-модифицированный
<400> 1146
atggatactt ggcttgtgtg ctgggccatc ttcagcctgc tgaaggccgg actgacagag 60
cccgaagtga cacagacacc cagccaccaa gtgacccaga tgggccaaga agtgatcctg 120
cgctgcgtgc ccatcagcaa ccacctgtac ttctactggt acagacagat cctgggccag 180
aaagtggaat tcctggtgtc cttctacaac aacgagatca gcgagaagtc cgagatcttc 240
gacgaccagt tcagcgtgga aagacccgac ggcagcaact tcaccctgaa gatcagaagc 300
accaagctcg aggacagcgc catgtacttt tgcgccagca cacccagaga cagaggcaaa 360
gaggccttct tcggccaagg caccagactg acagtggtc 399
<210> 1147
<211> 492
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 40 альфа, кодон-оптимизированный/ цистеин-модифицированный
<400> 1147
atgcacacca gcaccttcca gaacagaccc cagctgttcc tgctgatctg gaagaaactg 60
gtgcccggca atcccttcag gcggtcttgg atgaagcgcg agagagagat gctgctgatc 120
acctccatgc tggtgctgtg gatgcagctg agccaagtga acggccagca agtgatgcag 180
atccctcagt accagcacgt gcaagaaggc gaggacttca ccacctactg caacagcagc 240
accacactga gcaacatcca gtggtacaag cagcggcctg gcggacaccc tgtgtttctg 300
atccagctgg tcaagtccgg cgaagtgaag aagcagaagc ggctgacctt ccagttcggc 360
gaggccaaga agaacagcag cctgcacatc accgccacac agaccaccga tgtgggcacc 420
tacttttgcg ccggcagaaa caacttcaac aagttctact tcggcagcgg caccaagctg 480
aacgtgaagc cc 492
<210> 1148
<211> 396
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 40 бета, кодон-оптимизированный/ цистеин-модифицированный
<400> 1148
atggatactt ggcttgtgtg ctgggccatc ttcagcctgc tgaaggccgg actgacagag 60
cccgaagtga cacagacacc cagccaccaa gtgacccaga tgggccaaga agtgatcctg 120
cgctgcgtgc ccatcagcaa ccacctgtac ttctactggt acagacagat cctgggccag 180
aaagtggaat tcctggtgtc cttctacaac aacgagatca gcgagaagtc cgagatcttc 240
gacgaccagt tcagcgtgga aagacccgac ggcagcaact tcaccctgaa gatcagaagc 300
accaagctcg aggacagcgc catgtacttt tgcgccatca ccgccagaag cagctacgag 360
cagtatttcg gccctggcac cagactgacc gtgaca 396
<210> 1149
<211> 402
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 41 альфа, кодон-оптимизированный/ цистеин-модифицированный
<400> 1149
atgatgatca gcctgcgggt gctgctggtc atcctgtggc tgcaactgag ctgggtctgg 60
tcccagcgga aagaggtgga acaggacccc ggacctttca atgtgcctga aggcgccacc 120
gtggccttca actgcaccta cagcaatagc gccagccaga gcttcttctg gtacagacag 180
gactgccgga aagaacccaa gctgctgatg agcgtgtaca gcagcggcaa cgaggacggc 240
agattcacag cccagctgaa cagagccagc cagtacatct ccctgctgat ccgggatagc 300
aagctgagcg atagcgccac ctacctgtgc gtggtcacat ttgctctgac cggcggcttc 360
aagaccatct tcggagccgg caccagactg ttcgtgaagg cc 402
<210> 1150
<211> 402
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 41 бета, кодон-оптимизированный/ цистеин-модифицированный
<400> 1150
atggatactt ggcttgtgtg ctgggccatc ttcagcctgc tgaaggccgg actgacagag 60
cccgaagtga cacagacacc cagccaccaa gtgacccaga tgggccaaga agtgatcctg 120
cgctgcgtgc ccatcagcaa ccacctgtac ttctactggt acagacagat cctgggccag 180
aaagtggaat tcctggtgtc cttctacaac aacgagatca gcgagaagtc cgagatcttc 240
gacgaccagt tcagcgtgga aagacccgac ggcagcaact tcaccctgaa gatcagaagc 300
accaagctcg aggacagcgc catgtacttt tgcgccagca atccccggga cagagtgtcc 360
tacgagcagt actttggccc tggcaccaga ctgaccgtga ca 402
<210> 1151
<211> 492
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 42 альфа, кодон-оптимизированный/ цистеин-модифицированный
<400> 1151
atgcacacca gcaccttcca gaacagaccc cagctgttcc tgctgatctg gaagaaactg 60
gtgcccggca atcccttcag gcggtcttgg atgaagcgcg agagagagat gctgctgatc 120
acctccatgc tggtgctgtg gatgcagctg agccaagtga acggccagca agtgatgcag 180
atccctcagt accagcacgt gcaagaaggc gaggacttca ccacctactg caacagcagc 240
accacactga gcaacatcca gtggtacaag cagcggcctg gcggacaccc tgtgtttctg 300
atccagctgg tcaagtccgg cgaagtgaag aagcagaagc ggctgacctt ccagttcggc 360
gaggccaaga agaacagcag cctgcacatc accgccacac agaccaccga tgtgggcacc 420
tacttttgcg ccggacctga caacttcaac aagttctact tcggcagcgg caccaagctg 480
aacgtgaagc cc 492
<210> 1152
<211> 396
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 42 бета, кодон-оптимизированный/ цистеин-модифицированный
<400> 1152
atggatactt ggcttgtgtg ctgggccatc ttcagcctgc tgaaggccgg actgacagag 60
cccgaagtga cacagacacc cagccaccaa gtgacccaga tgggccaaga agtgatcctg 120
cgctgcgtgc ccatcagcaa ccacctgtac ttctactggt acagacagat cctgggccag 180
aaagtggaat tcctggtgtc cttctacaac aacgagatca gcgagaagtc cgagatcttc 240
gacgaccagt tcagcgtgga aagacccgac ggcagcaact tcaccctgaa gatcagaagc 300
accaagctcg aggacagcgc catgtacttt tgcgccaaga ccagcagaag cagctacgag 360
cagtacttcg gccctggcac cagactgaca gtgaca 396
<210> 1153
<211> 405
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 43 альфа, кодон-оптимизированный/ цистеин-модифицированный
<400> 1153
atgatgaagt ccctgcgggt gctgctggtc atcctgtggc tgcaactgag ctgggtctgg 60
tcccagcaga aagaggtgga acagaacagc ggccctctgt ctgttcctga aggcgctatc 120
gccagcctga actgcaccta cagcgataga ggcagccaga gcttcttctg gtacagacag 180
tacagcggca agagccccga gctgatcatg ttcatctaca gcaacggcga caaagaggac 240
ggccggttta cagcccagct gaacaaggcc agccagtacg tgtccctgct gatcagagat 300
agccagccta gcgacagcgc cacctatctg tgtgccgtga atgtgcctac cagcggcacc 360
tacaagtaca tcttcggcac cggcaccaga ctgaaggtgc tggct 405
<210> 1154
<211> 396
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 43 бета, кодон-оптимизированный/ цистеин-модифицированный
<400> 1154
atggatactt ggcttgtgtg ctgggccatc ttcagcctgc tgaaggccgg actgacagag 60
cccgaagtga cacagacacc cagccaccaa gtgacccaga tgggccaaga agtgatcctg 120
cgctgcgtgc ccatcagcaa ccacctgtac ttctactggt acagacagat cctgggccag 180
aaagtggaat tcctggtgtc cttctacaac aacgagatca gcgagaagtc cgagatcttc 240
gacgaccagt tcagcgtgga aagacccgac ggcagcaact tcaccctgaa gatcagaagc 300
accaagctcg aggacagcgc catgtacttt tgcgccagca cacccagaga cagctacgag 360
cagtatttcg gccctggcac cagactgacc gtgaca 396
<210> 1155
<211> 432
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 44 альфа, кодон-оптимизированный/ цистеин-модифицированный
<400> 1155
atggctcaag agctgggcat gcagtgtcag gccagaggaa tcctgcagca gatgtgggga 60
gtgttcctgc tgtacgtgtc catgaagatg ggcggcacca ccggccagaa catcgatcag 120
cctacagaga tgaccgccac cgagggcgcc atcgtgcaga tcaattgcac ctaccagacc 180
agcggcttca acggcctgtt ctggtatcag cagcatgccg gcgaggcccc taccttcctg 240
agctacaatg tgctggacgg cctggaagag aagggcagat tcagcagctt cctgtccaga 300
agcaagggct acagctacct gctgctgaaa gaactgcaga tgaaggacag cgcctcctac 360
ctgtgtgccc agtactctgg cggctaccag aaagtgacct ttggcaccgg caccaagctg 420
caagtgatcc cc 432
<210> 1156
<211> 387
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 44 бета, кодон-оптимизированный/ цистеин-модифицированный
<400> 1156
atgggcttta gactgctgtg ctgcgtggcc ttctgtctgc ttggagctgg ccctgtggat 60
agcggcgtta cccagacacc taagcacctg atcacagcca caggccagcg cgtgaccctg 120
agatgttctc ctagaagcgg cgacctgagc gtgtactggt atcagcagtc tctggaccag 180
ggcctgcagt tcctgatcca gtactacaac ggcgaggaaa gagccaaggg caacatcctg 240
gaacggttca gcgcccagca gttcccagat ctgcacagcg agctgaacct gagcagcctg 300
gaactgggag atagcgccct gtacttctgt gccagcagcg gcacacccga cacacagtat 360
tttggccctg gcaccagact gaccgtg 387
<210> 1157
<211> 498
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 45 альфа, кодон-оптимизированный/ цистеин-модифицированный
<400> 1157
atgcacacca gcaccttcca gaacagaccc cagctgttcc tgctgatctg gaagaaactg 60
gtgcccggca atcccttcag gcggtcttgg atgaagcgcg agagagagat gctgctgatc 120
acctccatgc tggtgctgtg gatgcagctg agccaagtga acggccagca agtgatgcag 180
atccctcagt accagcacgt gcaagaaggc gaggacttca ccacctactg caacagcagc 240
accacactga gcaacatcca gtggtacaag cagcggcctg gcggacaccc tgtgtttctg 300
atccagctgg tcaagtccgg cgaagtgaag aagcagaagc ggctgacctt ccagttcggc 360
gaggccaaga agaacagcag cctgcacatc accgccacac agaccaccga tgtgggcacc 420
tatttttgcg ccggatcttc tggcgccgga agctaccagc tgacatttgg caagggcacc 480
aagctgagcg tgatcccc 498
<210> 1158
<211> 402
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 45 бета, кодон-оптимизированный/ цистеин-модифицированный
<400> 1158
atgggaacca gactgctgtt ctgggtcgcc ttctgtctgc tgggcgccta tcatacaggt 60
gccggtgtct ctcagagccc cagcaacaaa gtgaccgaga agggcaaaga cgtggaactg 120
agatgcgacc ccatcagcgg acacacagcc ctgtactggt acagacagag actcggccag 180
ggcctcgagt tcctgatcta cttccaaggc aacagcgccc ctgacaagag cggcctgcct 240
agcgatagat tcagcgccga gagaacaggc gagagcgtgt ccacactgac catccagaga 300
acccagcaag aggacagcgc cgtgtacctg tgtgccagct ctctgtacct gggaaccacc 360
ggcgagctgt tctttggcga gggctctaga ctgaccgtgc tg 402
<210> 1159
<211> 399
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 46 альфа, кодон-оптимизированный/ цистеин-модифицированный
<400> 1159
atgctgctgc ttctggtgcc cgccttccaa gtgatcttta ccctcggcgg aacacgggcc 60
cagtctgtga cacagctgga ttctcaggtg cccgtgtttg aggaagcccc tgtcgagctg 120
agatgcaact acagcagcag cgtgtccgtg tacctgtttt ggtacgtgca gtaccccaac 180
cagggcctgc agctgctgct caagtatctg agcggcagca cactggtcaa gggcatcaat 240
ggcttcgagg ccgagttcaa caagagccag accagcttcc atctgcggaa gcccagcgtg 300
cacatcagcg ataccgccga gtacttctgc gccgtgtctc ctagcagcgg cacctacaag 360
tacatcttcg gcaccggcac cagactgaag gtgctggct 399
<210> 1160
<211> 402
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 46 бета, кодон-оптимизированный/ цистеин-модифицированный
<400> 1160
atgggaacaa gactgctgtg ttgggccgct ctgtgtctgc tgggagctga tcatacaggc 60
gccggtgtca gccagacacc tagcaacaaa gtgaccgaga agggcaaata cgtggaactg 120
agatgcgacc ccatcagcgg acacacagcc ctgtactggt acagacagtc tctcggccag 180
ggacctgagt tcctgatcta cttccaaggc accggcgctg ccgatgatag cggcctgcct 240
aacgatagat tcttcgccgt cagacccgag ggcagcgtgt ccacactgaa gatccagaga 300
accgagaggg gcgacagcgc cgtgtatctg tgtgccagct ctctgtatct cggcggcagc 360
gagacacagt actttggccc tggtacacgg ctgctggttc tg 402
<210> 1161
<211> 405
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 47 альфа, кодон-оптимизированный/ цистеин-модифицированный
<400> 1161
atgatgaagt ccctgcgggt gctgctggtc atcctgtggc tgcaactgag ctgggtctgg 60
tcccagcaga aagaggtgga acagaacagc ggccctctgt ctgttcctga aggcgctatc 120
gccagcctga actgcaccta cagcgataga ggcagccaga gcttcttctg gtacagacag 180
tacagcggca agagccccga gctgatcatg ttcatctaca gcaacggcga caaagaggac 240
ggccggttta cagcccagct gaacaaggcc agccagtacg tgtccctgct gatcagagat 300
agccagccta gcgacagcgc cacctatctg tgtgccgtga atctgctgag cggcagcgct 360
agacagctga catttggctc tggcacccag ctgaccgtgc tgcct 405
<210> 1162
<211> 396
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 47 бета, кодон-оптимизированный/ цистеин-модифицированный
<400> 1162
atggatactt ggcttgtgtg ctgggccatc ttcagcctgc tgaaggccgg actgacagag 60
cccgaagtga cacagacacc cagccaccaa gtgacccaga tgggccaaga agtgatcctg 120
cgctgcgtgc ccatcagcaa ccacctgtac ttctactggt acagacagat cctgggccag 180
aaagtggaat tcctggtgtc cttctacaac aacgagatca gcgagaagtc cgagatcttc 240
gacgaccagt tcagcgtgga aagacccgac ggcagcaact tcaccctgaa gatcagaagc 300
accaagctcg aggacagcgc catgtacttt tgcgccatga ccggcagaac cacctacgag 360
cagtatttcg gccctggcac cagactgacc gtgaca 396
<210> 1163
<211> 399
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 48 альфа, кодон-оптимизированный/ цистеин-модифицированный
<400> 1163
atgatgaagt ccctgcgggt gctgctggtc atcctgtggc tgcaactgag ctgggtctgg 60
tcccagcaga aagaggtgga acaggaccct ggacctctgt ctgttcctga gggcgccatc 120
gtgtccctga actgcaccta cagcaacagc gccttccagt acttcatgtg gtacagacag 180
tacagccgga agggccccga gctgctgatg tacacataca gcagcggcaa caaagaggac 240
ggccggttta cagcccaggt ggacaagagc agcaagtaca tctccctgtt catccgggac 300
agccagccta gcgatagcgc cacatatctg tgcgccatga gaatccaggg cgcccagaaa 360
ctggtgttcg gccagggaac cagactgacc atcaatccc 399
<210> 1164
<211> 396
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 48 бета, кодон-оптимизированный/ цистеин-модифицированный
<400> 1164
atggatactt ggcttgtgtg ctgggccatc ttcagcctgc tgaaggccgg actgacagag 60
cccgaagtga cacagacacc cagccaccaa gtgacccaga tgggccaaga agtgatcctg 120
cgctgcgtgc ccatcagcaa ccacctgtac ttctactggt acagacagat cctgggccag 180
aaagtggaat tcctggtgtc cttctacaac aacgagatca gcgagaagtc cgagatcttc 240
gacgaccagt tcagcgtgga aagacccgac ggcagcaact tcaccctgaa gatcagaagc 300
accaagctcg aggacagcgc catgtacttt tgcgccagca ccggcagagt gtcctacgag 360
cagtatttcg gccctggcac cagactgacc gtgaca 396
<210> 1165
<211> 405
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 49 альфа, кодон-оптимизированный/ цистеин-модифицированный
<400> 1165
atgatgaagt ccctgcgggt gctgctggtc atcctgtggc tgcaactgag ctgggtctgg 60
tcccagcaga aagaggtgga acagaacagc ggccctctgt ctgttcctga aggcgctatc 120
gccagcctga actgcaccta cagcgataga ggcagccaga gcttcttctg gtacagacag 180
tacagcggca agagccccga gctgatcatg ttcatctaca gcaacggcga caaagaggac 240
ggccggttta cagcccagct gaacaaggcc agccagtacg tgtccctgct gatcagagat 300
agccagccta gcgacagcgc cacctatctg tgcgccgtga atatcggcac cagcggcacc 360
tacaagtaca tcttcggcac cggcaccaga ctgaaggtgc tggct 405
<210> 1166
<211> 396
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 49 бета, кодон-оптимизированный/ цистеин-модифицированный
<400> 1166
atggatacct ggctcgtgtg ctgggccatc ttcagcctgc tgaaagccgg actgaccgag 60
cctgaagtga cccagacacc tagccaccaa gtgacacaga tgggccaaga agtgatcctg 120
cgctgcgtgc ccatcagcaa ccacctgtac ttctactggt acagacagat cctgggccag 180
aaagtggaat tcctggtgtc cttctacaac aacgagatca gcgagaagtc cgagatcttc 240
gacgaccagt tcagcgtgga aagacccgac ggcagcaact tcaccctgaa gatcagaagc 300
accaagctcg aggacagcgc catgtacttt tgcgccagca caccccggta cagctacgag 360
cagtatttcg gccctggcac cagactgacc gtgaca 396
<210> 1167
<211> 396
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 50 альфа, кодон-оптимизированный/ цистеин-модифицированный
<400> 1167
atgaagacct tcgccggctt cagcttcctg ttcctgtggc tgcagctgga ctgcatgagc 60
agaggcgagg atgtggaaca gagcctgttt ctgagcgtcc gcgagggcga tagcagcgtg 120
atcaattgca cctacaccga cagcagcagc acctacctgt actggtacaa gcaagagcct 180
ggcgccggac tgcagctgct gacctacatc ttcagcaaca tggacatgaa gcaggaccag 240
cggctgaccg tgctgctgaa caagaaggac aagcacctga gcctgcggat cgccgataca 300
cagacaggcg acagcgccat ctacttttgc gccgagtctc ctccaggcac ctacaagtac 360
atcttcggca ccggcaccag actgaaggtg ctggct 396
<210> 1168
<211> 396
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 50 бета, кодон-оптимизированный/ цистеин-модифицированный
<400> 1168
atgggcttca gactgctgtg ctgcgtggcc ttttgtctgc ttggagccgg acctgtggat 60
agcggcgtta cccagacacc taagcacctg atcacagcca caggccagcg cgtgaccctg 120
agatgttctc ctagaagcgg cgacctgagc gtgtactggt atcagcagtc tctggaccag 180
ggcctgcagt tcctgatcca ctactacaac ggcgaggaaa gagccaaggg caacatcctg 240
gaacggttca gcgcccagca gttcccagat ctgcacagcg agctgaacct gagcagcctg 300
gaactgggag atagcgccct gtacttctgt gccagcagcg ccacaagaga tgcctacggc 360
tacacatttg gcagcggcac cagactgaca gtggtg 396
<210> 1169
<211> 408
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 51 альфа, кодон-оптимизированный/ цистеин-модифицированный
<400> 1169
atgatgaagt ccctgcgggt gctgctggtc atcctgtggc tgcaactgag ctgggtctgg 60
tcccagcaga aagaggtgga acagaacagc ggccctctgt ctgttcctga aggcgctatc 120
gccagcctga actgcaccta cagcgataga ggcagccaga gcttcttctg gtacagacag 180
tacagcggca agagccccga gctgatcatg agcatctaca gcaacggcga caaagaggac 240
ggccggttta cagcccagct gaacaaggcc agccagtacg tgtccctgct gatcagagat 300
agccagccta gcgacagcgc cacctatctg tgcgccgtga atatgagagg cggcggaagc 360
aactacaagc tgacctttgg caagggcacc ctgctgaccg tgaatccc 408
<210> 1170
<211> 396
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 51 бета, кодон-оптимизированный/ цистеин-модифицированный
<400> 1170
atggatacct ggctcgtgtg ctgggccatc ttcagcctgc tgaaagccgg actgaccgag 60
cctgaagtga cccagacacc tagccaccaa gtgacacaga tgggccaaga agtgatcctg 120
cgctgcgtgc ccatcagcaa ccacctgtac ttctactggt acagacagat cctgggccag 180
aaagtggaat tcctggtgtc cttctacaac aacgagatca gcgagaagtc cgagatcttc 240
gacgaccagt tcagcgtgga aagacccgac ggcagcaact tcaccctgaa gatcagaagc 300
accaagctcg aggacagcgc catgtacttt tgcgccattg ccagccgggt gtcctacgag 360
cagtatttcg gccctggcac cagactgacc gtgaca 396
<210> 1171
<211> 381
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 52 альфа, кодон-оптимизированный/ цистеин-модифицированный
<400> 1171
atggtgctga agttcagcgt gtccatcctg tggatccagc tggcctgggt ttccacacag 60
ctgctggaac agagccctca gttcctgagc atccaagagg gcgagaacct gaccgtgtac 120
tgcaacagca gcagcgtgtt cagcagcctg cagtggtaca gacaagagcc tggcgaagga 180
cctgtgctgc tggtcacagt tgtgacaggc ggcgaagtga agaagctgaa gcggctgacc 240
ttccagttcg gcgacgccag aaaggatagc tccctgcaca ttaccgctgc tcagccaggc 300
gataccggcc tgtatctttg tgccggcgct cgggattaca agctgtcttt tggcgccgga 360
accaccgtga cagtgcgggc t 381
<210> 1172
<211> 396
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 52 бета, кодон-оптимизированный/ цистеин-модифицированный
<400> 1172
atgggcttca gactgctgtg ctgcgtggcc ttttgtctgc ttggagccgg acctgtggat 60
agcggcgtta cccagacacc taagcacctg atcacagcca caggccagcg cgtgaccctg 120
agatgttctc ctagaagcgg cgacctgagc gtgtactggt atcagcagtc tctggaccag 180
ggcctgcagt tcctgatcca gtactacaac ggcgaggaaa gagccaaggg caacatcctg 240
gaacggttca gcgcccagca gttcccagat ctgcacagcg agctgaacct gagcagcctg 300
gaactgggag atagcgccct gtacttctgt gccagctctg tgggcgatct gaacaacgag 360
cagttcttcg gccctggcac cagactgaca gttctg 396
<210> 1173
<211> 402
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 53 альфа, кодон-оптимизированный/ цистеин-модифицированный
<400> 1173
atggccagcg ctcctatctc tatgctggcc atgctgttca ccctgagcgg actgagagca 60
cagtctgtgg cccagcctga ggaccaagtg aatgtggccg agggcaatcc cctgaccgtg 120
aagtgtacct acagcgtgtc cggcaatccc tacctgtttt ggtacgtgca gtaccccaac 180
cggggcctgc agttcctgct gaagtacatc accggcgaca acctggtcaa gggcagctat 240
ggattcgagg ccgagttcaa caagagccag accagcttcc acctgaagaa acccagcgct 300
ctggtgtccg atagcgccct gtatttctgc gccgtcagag attttggcag cggcacctac 360
aagtacatct tcggcaccgg caccagactg aaggtgctgg ct 402
<210> 1174
<211> 404
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 53 бета, кодон-оптимизированный/ цистеин-модифицированный
<400> 1174
atgggcacaa gcctgctgtg ttgggtcgtg ctgggctttc tgggcacaga tcatacaggc 60
gccggtgtca gccagtctcc tcggtacaaa gtgaccaagc gcggacagga tgtggccctg 120
agatgcgatc ctatctctgg ccacgtgtcc ctgtactggt acagacaggc tctcggacag 180
ggccccgagt tcctgaccta cttcaattac gaggcccagc aggacaagag cggcctgcct 240
aacgatagat tcagcgccga aagacccgag ggcagcatca gcacactgac catccagaga 300
accgagcaga gggacagcgc catgtacaga tgtgcctcta gcggcagcgg caccagcggc 360
tacaatgagc agtttttcgg ccctggcacc agactgacag tgct 404
<210> 1175
<211> 402
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 54 альфа, кодон-оптимизированный/ цистеин-модифицированный
<400> 1175
atgaagaagc acctgaccac ctttctggtc atcctgtggc tgtacttcta cagaggcaac 60
ggcaagaacc aggtggaaca gagccctcag agcctgatca tcctggaagg caagaactgc 120
accctgcagt gcaactacac cgtgtctccc ttcagcaacc tgcggtggta caagcaggat 180
acaggcagag gccctgtgtc tctgaccatc atgaccttca gcgagaacac caagagcaac 240
ggcagataca ccgccacact ggacgccgat acaaagcaga gcagcctgca catcacagcc 300
agccagctga gcgatagcgc cagctacatc tgcgtggtgt ccagagacaa ctacggccag 360
aacttcgtgt tcggccctgg cacaagactg agcgtgctgc ct 402
<210> 1176
<211> 396
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 54 бета, кодон-оптимизированный/ цистеин-модифицированный
<400> 1176
atggatactt ggctcgtgtg ctgggccatc ttcagcctgc tgaaagccgg actgaccgag 60
cctgaagtga cacagacacc tagccaccaa gtgacccaga tgggccaaga ggtcatactg 120
agatgcgtgc ccatcagcaa ccacctgtac ttctactggt acagacagat cctgggccag 180
aaggtggaat tcctcgtcag cttctacaac aacgagatca gcgagaagtc cgagatcttc 240
gacgaccagt tcagcgtgga aagacccgac ggcagcaact tcaccctgaa gatcagaagc 300
accaagctcg aggacagcgc catgtacttt tgcgccatca ccgaccggac caactacggc 360
tacacatttg gcagcggcac cagactgaca gtggtc 396
<210> 1177
<211> 396
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 55 альфа, кодон-оптимизированный/ цистеин-модифицированный
<400> 1177
atgaagacct tcgccggctt cagcttcctg ttcctgtggc tgcagctgga ctgtatgagc 60
agaggcgagg atgtggaaca gagcctgttt ctgagcgtca gagaaggcga cagcagcgtg 120
atcaactgca cctacaccga cagctccagc acctacctgt actggtacaa gcaagaacct 180
ggcgccggac tgcagctgct gacctacatc ttcagcaaca tggacatgaa gcaggaccag 240
cggctgaccg tgctgctgaa caagaaggat aagcacctga gcctgcggat cgccgataca 300
cagacaggcg actccgccat ctacttttgt gctgtgccta gcggcgccac caacaagctg 360
atctttggaa caggcacact gctggccgtg cagccc 396
<210> 1178
<211> 396
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 55 бета, кодон-оптимизированный/ цистеин-модифицированный
<400> 1178
atggatactt ggctcgtgtg ctgggccatc ttcagcctgc tgaaagccgg actgaccgag 60
cctgaagtga cacagacacc tagccaccaa gtgacccaga tgggccaaga ggtcatactg 120
agatgcgtgc ccatcagcaa ccacctgtac ttctactggt acagacagat cctgggccag 180
aaggtggaat tcctcgtcag cttctacaac aacgagatca gcgagaagtc cgagatcttc 240
gacgaccagt tcagcgtgga aagacccgac ggcagcaact tcaccctgaa gatcagaagc 300
accaagctcg aggacagcgc catgtacttt tgcgccagca ccaccagaag cagctacgag 360
cagtatttcg gccctggcac cagactgacc gtgaca 396
<210> 1179
<211> 66
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCR 15/ TCR 16/ TCR 17/ TCR 18/ TCR 19/ TCR 20/
TCR 21/ TCR22/ TCR 23/ TCR 24/ TCR 25/ TCR 26/ TCR
27/ TCR 28/ TCR 29/ TCR 30/ TCR 31/ TCR 32/ TCR
33/ TCR 34 P2A
<400> 1179
ggctctggcg ccaccaactt tagcctgctg aagcaggccg gcgatgtgga ggagaatcct 60
ggccca 66
<210> 1180
<211> 66
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCR 35/ TCR 36/ TCR 38/ TCR 40/ TCR 41/ TCR 42/
TCR 43/ TCR 44/ TCR 45/ TCR 46/ TCR 47/ TCR 48 P2A
<400> 1180
ggaagcggcg ccacaaactt ctcactgctg aaacaggctg gcgacgtgga ggagaatcct 60
ggccca 66
<210> 1181
<211> 66
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCR 37/ TCR 39 P2A
<400> 1181
ggcagcggcg ccaccaactt cagcctgctt aaacaagctg gcgacgtgga ggagaatcct 60
ggccca 66
<210> 1182
<211> 20
<212> ДНК
<213> Homo sapiens
<220>
<223> TRAC последовательность-мишень
<400> 1182
attcaccgat tttgattctc 20
<210> 1183
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR альфа E7(11-19) CDR3 консенсус
<220>
<221> ВАРИАНТ
<222> (1)...(1)
<223> X1 представляет собой V или A
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 представляет собой V, A, G, Q, M или E
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 представляет собой S, G, A, N,Y, R,T или P
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 представляет собой E, A, S, G, R. F, N, D, V, P, L, I или M
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 представляет собой R, N, H, T, D, G, S, A, P, L, Q или F
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 представляет собой G, H, N, A, S, L, T или отсутствует
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 представляет собой T, S, G или отсутствует
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X8 представляет собой G или отсутствует
<220>
<221> ВАРИАНТ
<222> (9)...(9)
<223> X9 представляет собой G, Y, N, S или отсутствует
<220>
<221> ВАРИАНТ
<222> (10)...(10)
<223> X10 представляет собой T, G, S, D, F, Y, A, N или отсутствует
<220>
<221> ВАРИАНТ
<222> (11)...(11)
<223> X11 представляет собой Y, F, Y, Q, N или R
<220>
<221> ВАРИАНТ
<222> (12)...(12)
<223> X12 представляет собой N, K, Q или D
<220>
<221> ВАРИАНТ
<222> (13)...(13)
<223> X13 представляет собой Y, L, T, F, M или V
<220>
<221> ВАРИАНТ
<222> (14)...(14)
<223> X14 представляет собой I, T, S, V, R или Y
<400> 1183
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5 10
<210> 1184
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR альфа E7(11-19) CDR3 консенсус
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 представляет собой S, N или T
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 представляет собой R или F
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 представляет собой D или A
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 представляет собой N или L
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7is T или отсутствует
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X8 представляет собой Y или G
<220>
<221> ВАРИАНТ
<222> (10)...(10)
<223> X10 представляет собой Q или F
<220>
<221> ВАРИАНТ
<222> (11)...(11)
<223> X11 представляет собой N или K
<220>
<221> ВАРИАНТ
<222> (12)...(12)
<223> X12 представляет собой F или T
<220>
<221> ВАРИАНТ
<222> (13)...(13)
<223> X13 представляет собой V или I
<400> 1184
Val Val Xaa Xaa Xaa Xaa Xaa Xaa Gly Xaa Xaa Xaa Xaa
1 5 10
<210> 1185
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR альфа E7(11-19) CDR3 консенсус
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 представляет собой A, G, V, Q, M или E
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 представляет собой S, G, N, A, Y, R или P
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 представляет собой E, S, A, G, F, N, D, V, P, L, I, M или R
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 представляет собой R, N, H, T, D, G, S, P, L, Q или F
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 представляет собой G, H, A, S, T или отсутствует
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 представляет собой T, S, G или отсутствует
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X8 представляет собой G или отсутствует
<220>
<221> ВАРИАНТ
<222> (9)...(9)
<223> X9 представляет собой G, N, S или отсутствует
<220>
<221> ВАРИАНТ
<222> (10)...(10)
<223> X10 представляет собой T, G, S, D, F, Y, A или N
<220>
<221> ВАРИАНТ
<222> (11)...(11)
<223> X11 представляет собой Y, F, Q, R или N
<220>
<221> ВАРИАНТ
<222> (12)...(12)
<223> X12 представляет собой K, Q или D
<220>
<221> ВАРИАНТ
<222> (13)...(13)
<223> X13 представляет собой Y, L, T, M, F или V
<220>
<221> ВАРИАНТ
<222> (14)...(14)
<223> X14 представляет собой I, T, S, R, Y или V
<400> 1185
Ala Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5 10
<210> 1186
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR альфа E7(11-19) CDR3 консенсус
<220>
<221> ВАРИАНТ
<222> (1)...(1)
<223> X1 представляет собой A или V
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 представляет собой A, V или E
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 представляет собой S, N, T, R или P
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 представляет собой E, A, G, F, V, P, I, D или S
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 представляет собой R, H, T, A P, S, G или F
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 представляет собой G, H, L, T, S, A или отсутствует
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 представляет собой S, T или отсутствует
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X8 представляет собой G или отсутствует
<220>
<221> ВАРИАНТ
<222> (9)...(9)
<223> X9 представляет собой G, T или отсутствует
<220>
<221> ВАРИАНТ
<222> (10)...(10)
<223> X10 представляет собой F, Y или N
<220>
<221> ВАРИАНТ
<222> (12)...(12)
<223> X12 представляет собой Y, T, L
<400> 1186
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Lys Xaa Ile
1 5 10
<210> 1187
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR альфа E7(11-19) CDR3 консенсус
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 представляет собой A, V или E
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 представляет собой S, N или R
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 представляет собой E, G, V, P, I или D
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 представляет собой R, T, P, S, G или F
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 представляет собой G, T, S или отсутствует
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7is S или отсутствует
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X8 представляет собой G или отсутствует
<220>
<221> ВАРИАНТ
<222> (9)...(9)
<223> X9 представляет собой T или отсутствует
<400> 1187
Ala Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Tyr Lys Tyr Ile
1 5 10
<210> 1188
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR альфа E7(11-19) CDR3 консенсус
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 представляет собой G, V, Q или M
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 представляет собой G, A, Y, S, N или R
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 представляет собой S, G, L, I, M или R
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 представляет собой N, D, G, S, L, Q или R
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 представляет собой A, S, G или отсутствует
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 представляет собой G или отсутствует
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X8 представляет собой G или отсутствует
<220>
<221> ВАРИАНТ
<222> (9)...(9)
<223> X9 представляет собой G, N, S или отсутствует
<220>
<221> ВАРИАНТ
<222> (10)...(10)
<223> X10 представляет собой S, D, Y, A, N или отсутствует
<220>
<221> ВАРИАНТ
<222> (11)...(11)
<223> X11 представляет собой Y, Q или R
<220>
<221> ВАРИАНТ
<222> (12)...(12)
<223> X12 представляет собой K или Q
<220>
<221> ВАРИАНТ
<222> (13)...(13)
<223> X13 представляет собой L или V
<220>
<221> ВАРИАНТ
<222> (14)...(14)
<223> X14 представляет собой S, T или V
<400> 1188
Ala Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5 10
<210> 1189
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR альфа E7(11-19) CDR3 консенсус
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 представляет собой G, V или Q
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 представляет собой G, Y, S или N
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 представляет собой S, L, или M
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 представляет собой N, G, L или R
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 представляет собой A, S, G или отсутствует
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 представляет собой G или отсутствует
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X8 представляет собой G или отсутствует
<220>
<221> ВАРИАНТ
<222> (9)...(9)
<223> X9 представляет собой G, S или отсутствует
<220>
<221> ВАРИАНТ
<222> (10)...(10)
<223> X10 представляет собой S, Y, A, N или отсутствует
<220>
<221> ВАРИАНТ
<222> (11)...(11)
<223> X11 представляет собой Y, Q или R
<220>
<221> ВАРИАНТ
<222> (12)...(12)
<223> X12 представляет собой K или Q
<220>
<221> ВАРИАНТ
<222> (13)...(13)
<223> X13 представляет собой L или V
<400> 1189
Ala Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Thr
1 5 10
<210> 1190
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR альфа E7(11-19) CDR3 консенсус
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 представляет собой G или V
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 представляет собой A или Y
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 представляет собой G, S или R
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 представляет собой D или S
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 представляет собой N или отсутствует
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 представляет собой D или отсутствует
<400> 1190
Ala Xaa Xaa Xaa Xaa Xaa Xaa Tyr Lys Leu Ser
1 5 10
<210> 1191
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR альфа E7(11-19) CDR1 консенсус
<220>
<221> ВАРИАНТ
<222> (1)...(1)
<223> X1 представляет собой N, S, D, T или V
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 представляет собой S, V, R, T или I
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 представляет собой M, F, G, S, N, A, L, V или P
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 представляет собой F, S, N, A или отсутствует
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 представляет собой D, S, Q, Y, N, V, T или P
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 представляет собой Y, S, R, N, G или T
<400> 1191
Xaa Xaa Xaa Xaa Xaa Xaa
1 5
<210> 1192
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR альфа E7(11-19) CDR2 консенсус
<220>
<221> ВАРИАНТ
<222> (1)...(1)
<223> X1 представляет собой I, V, L, G, , N, T, Y или M
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 представляет собой S, V, Y, L, P, F, I или T
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 представляет собой S, Y, K, L, T или F
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 представляет собой I, G, N, A, S или отсутствует
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 представляет собой S, D или отсутствует
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 представляет собой K, G, N, S, D, T или E
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 представляет собой D, E, G, A, K, L или N
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X8 представляет собой K, V, D, P, N, T, L или M
<400> 1192
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5
<210> 1193
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR бета E7(11-19) CDR3 консенсус
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 представляет собой S, M, I, K или V
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 представляет собой S, T, N или A
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 представляет собой R, V P, S, T, G, L, A, I или D
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 представляет собой F, G, R, Y, S, L, V или T
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 представляет собой L, G, D, A, S, T, V, R или отсутствует
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7is G, D, R, S, T или отсутствует
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X8 представляет собой S или отсутствует
<220>
<221> ВАРИАНТ
<222> (9)...(9)
<223> X9 представляет собой S, H, G, R, V, T, D, L или отсутствует
<220>
<221> ВАРИАНТ
<222> (10)...(10)
<223> X10 представляет собой T, S, A, Y, N, G или P
<220>
<221> ВАРИАНТ
<222> (11)...(11)
<223> X11 представляет собой D,Y, N, E, K или G
<220>
<221> ВАРИАНТ
<222> (12)...(12)
<223> X12 представляет собой T, E, G или K
<220>
<221> ВАРИАНТ
<222> (13)...(13)
<223> X13 представляет собой Q, Y, A или L
<220>
<221> ВАРИАНТ
<222> (14)...(14)
<223> X14 представляет собой Y, F, T или I
<400> 1193
Ala Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5 10
<210> 1194
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR бета E7(11-19) CDR3 консенсус
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 представляет собой S, M, I или K
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 представляет собой P, T, G, A, S или D
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 представляет собой R или S
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 представляет собой D, G, S, T или V
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 представляет собой R, S или отсутствует
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X8 представляет собой T, Y, G, N или S
<220>
<221> ВАРИАНТ
<222> (9)...(9)
<223> X9 isY, N или K
<220>
<221> ВАРИАНТ
<222> (10)...(10)
<223> X10 представляет собой E или G
<220>
<221> ВАРИАНТ
<222> (11)...(11)
<223> X11 представляет собой Q, A или Y
<220>
<221> ВАРИАНТ
<222> (12)...(12)
<223> X12 представляет собой Y, F или T
<400> 1194
Ala Xaa Thr Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5 10
<210> 1195
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR бета E7(11-19) CDR3 консенсус
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 представляет собой S, M, I или K
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 представляет собой S, T, A или N
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 представляет собой R, V, S, P, T, G, L или A
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 представляет собой F, G, R, Y, S, V или T
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 представляет собой L, G, D, A, S, T, V или отсутствует
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 представляет собой G, D, R, T или отсутствует
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X8 представляет собой S или отсутствует
<220>
<221> ВАРИАНТ
<222> (9)...(9)
<223> X9 представляет собой S, H, G, R, V, T, L или отсутствует
<220>
<221> ВАРИАНТ
<222> (10)...(10)
<223> X10 представляет собой T, S, Y, A, N, G или P
<220>
<221> ВАРИАНТ
<222> (11)...(11)
<223> X11 представляет собой D, Y, N, K, E или G
<220>
<221> ВАРИАНТ
<222> (12)...(12)
<223> X12 представляет собой T или E
<220>
<221> ВАРИАНТ
<222> (13)...(13)
<223> X13 представляет собой Q, A или L
<220>
<221> ВАРИАНТ
<222> (14)...(14)
<223> X14 представляет собой Y или F
<400> 1195
Ala Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5 10
<210> 1196
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR бета E7(11-19) CDR3 консенсус
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 представляет собой S, M, I или K
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 представляет собой S, T, A или N
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 представляет собой R, P, S, G, L, A или T
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 представляет собой F, R, Y, V или T
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 представляет собой L, D, A, S, T, V или отсутствует
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 представляет собой G, R или отсутствует
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X8 представляет собой S, G, V или отсутствует
<220>
<221> ВАРИАНТ
<222> (9)...(9)
<223> X9 представляет собой T, A, G, N, S или P
<220>
<221> ВАРИАНТ
<222> (10)...(10)
<223> X10 представляет собой D, Y или E
<220>
<221> ВАРИАНТ
<222> (11)...(11)
<223> X11 представляет собой T или E
<400> 1196
Ala Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Gln Tyr
1 5 10
<210> 1197
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR бета E7(11-19) CDR3 консенсус
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 представляет собой S, M, I или K
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 представляет собой S, T, A или N
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 представляет собой P, S, G, T, или A
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 представляет собой R или Y
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 представляет собой D, A, S, T или V
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 представляет собой R или отсутствует
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X8 представляет собой G, V или отсутствует
<220>
<221> ВАРИАНТ
<222> (9)...(9)
<223> X9 представляет собой S, T, A или N
<400> 1197
Ala Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Tyr Glu Gln Tyr
1 5 10
<210> 1198
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR бета E7(11-19) CDR3 консенсус
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 представляет собой T, P или G
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 представляет собой R или S
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 представляет собой S, D, G или V
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 представляет собой D или отсутствует
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X8 представляет собой S или отсутствует
<220>
<221> ВАРИАНТ
<222> (9)...(9)
<223> X9 представляет собой S, R или отсутствует
<220>
<221> ВАРИАНТ
<222> (10)...(10)
<223> X10 представляет собой S, T, Y или G
<220>
<221> ВАРИАНТ
<222> (11)...(11)
<223> X11 представляет собой Y, N или K
<220>
<221> ВАРИАНТ
<222> (13)...(13)
<223> X13 представляет собой Q или A
<220>
<221> ВАРИАНТ
<222> (14)...(14)
<223> X14 представляет собой Y или F
<400> 1198
Ala Ser Thr Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Glu Xaa Xaa
1 5 10
<210> 1199
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR бета E7(11-19) CDR3 консенсус
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 представляет собой S или I
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 представляет собой S или T
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 представляет собой L, A или D
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 представляет собой L, T или R
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 представляет собой L, T или R
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 представляет собой G, D или отсутствует
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X8 представляет собой A или N
<400> 1199
Ala Xaa Xaa Xaa Xaa Xaa Xaa Xaa Tyr Gly Tyr Thr
1 5 10
<210> 1200
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR бета E7(11-19) CDR3 консенсус
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 представляет собой S, V или I
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 представляет собой S, N или A
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 представляет собой R, V, S, L, P, G, I или A
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 представляет собой F, G, Y, L, V, R, T или S
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 представляет собой L, G, A, D, R, V или отсутствует
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 представляет собой G, D, R, S, T или отсутствует
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X8 представляет собой S или отсутствует
<220>
<221> ВАРИАНТ
<222> (9)...(9)
<223> X9 представляет собой S, H, G, V, T, D, L или отсутствует
<220>
<221> ВАРИАНТ
<222> (10)...(10)
<223> X10 представляет собой T, S, A, G, P, N или Y
<220>
<221> ВАРИАНТ
<222> (11)...(11)
<223> X11 представляет собой D, Y, E, G или N
<220>
<221> ВАРИАНТ
<222> (12)...(12)
<223> X12 представляет собой T, E, G или K
<220>
<221> ВАРИАНТ
<222> (13)...(13)
<223> X13 представляет собой Q, Y или L
<220>
<221> ВАРИАНТ
<222> (14)...(14)
<223> X14 представляет собой Y, F, T или I
<400> 1200
Ala Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5 10
<210> 1201
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR бета E7(11-19) CDR3 консенсус
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 представляет собой R, V, S, L, G или A
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 представляет собой F, G, Y, L, V, T или S
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 представляет собой A, L, R, D, G или отсутствует
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 представляет собой G, D, T или отсутствует
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X8 представляет собой S или отсутствует
<220>
<221> ВАРИАНТ
<222> (9)...(9)
<223> X9 представляет собой S, H, G, T, D, L или отсутствует
<220>
<221> ВАРИАНТ
<222> (10)...(10)
<223> X10 представляет собой T, S, A, G, P, N или Y
<220>
<221> ВАРИАНТ
<222> (11)...(11)
<223> X11 представляет собой D, Y, E, G или N
<220>
<221> ВАРИАНТ
<222> (12)...(12)
<223> X12 isT, E, G или T
<220>
<221> ВАРИАНТ
<222> (13)...(13)
<223> X13 представляет собой Q, Y или L
<220>
<221> ВАРИАНТ
<222> (14)...(14)
<223> X14 isY, F или T
<400> 1201
Ala Ser Ser Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5 10
<210> 1202
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR бета E7(11-19) CDR3 консенсус
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 представляет собой R, L или G
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 представляет собой F, V, T или Y
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 представляет собой L, A или отсутствует
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 представляет собой G или отсутствует
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X8 представляет собой S, G или отсутствует
<220>
<221> ВАРИАНТ
<222> (9)...(9)
<223> X9 представляет собой T, G, P или S
<220>
<221> ВАРИАНТ
<222> (10)...(10)
<223> X10 представляет собой D или E
<400> 1202
Ala Ser Ser Xaa Xaa Xaa Xaa Xaa Xaa Xaa Thr Gln Tyr
1 5 10
<210> 1203
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR бета E7(11-19) CDR1 консенсус
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 представляет собой G или N
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 представляет собой H или D
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 isT, L, N или V
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 представляет собой A, S, Y или T
<400> 1203
Ser Xaa Xaa Xaa Xaa
1 5
<210> 1204
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR бета E7(11-19) CDR2 консенсус
<220>
<221> ВАРИАНТ
<222> (1)...(1)
<223> X1 представляет собой F или Y
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 представляет собой Q, Y или N
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 представляет собой G, N, R или Y
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 представляет собой N, G, E или T
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 представляет собой S, E, A или G
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 представляет собой A, E, I или Q
<400> 1204
Xaa Xaa Xaa Xaa Xaa Xaa
1 5
<210> 1205
<211> 18
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR альфа E6(29-38) CDR3 консенсус
<220>
<221> ВАРИАНТ
<222> (1)...(1)
<223> X1 представляет собой A, I или V
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 представляет собой M, L, A, V, S или E
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 представляет собой R, L, N, S, Q, K, G или W
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 представляет собой E, V, P, T, F, A, G, N, D или L
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 представляет собой G, I, D, L, A, P, H, N, R, T или отсутствует
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 представляет собой G, N, R, T, M, S, P или отсутствует
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 представляет собой G, V, D, L, Q, T, R, N или отсутствует
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X8 представляет собой T, D, S, L, G или отсутствует
<220>
<221> ВАРИАНТ
<222> (9)...(9)
<223> X9 представляет собой A, G, Q или отсутствует
<220>
<221> ВАРИАНТ
<222> (10)...(10)
<223> X10 представляет собой G или отсутствует
<220>
<221> ВАРИАНТ
<222> (11)...(11)
<223> X11 представляет собой G или отсутствует
<220>
<221> ВАРИАНТ
<222> (12)...(12)
<223> X12 представляет собой T или отсутствует
<220>
<221> ВАРИАНТ
<222> (13)...(13)
<223> X13 представляет собой S, A, T, G или отсутствует
<220>
<221> ВАРИАНТ
<222> (14)...(14)
<223> X14 представляет собой G, Y, T, N, A, W или отсутствует
<220>
<221> ВАРИАНТ
<222> (15)...(15)
<223> X15 представляет собой F, G, N, T, Y, D, S, R, Q или E
<220>
<221> ВАРИАНТ
<222> (16)...(16)
<223> X16 представляет собой K, P, A, N, D или Q
<220>
<221> ВАРИАНТ
<222> (17)...(18)
<223> X17 представляет собой L, M, I, V или T
X18 представляет собой I, T, V, N, F, R или Q
<400> 1205
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5 10 15
Xaa Xaa
<210> 1206
<211> 18
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR альфа E6(29-38) CDR3 консенсус
<220>
<221> ВАРИАНТ
<222> (1)...(1)
<223> X1 представляет собой A, I или V
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 представляет собой L, M, V или E
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 представляет собой L, R, N, G или S
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 представляет собой V, T, F, N, E, P, G или L
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 представляет собой I, A, P, N, G или T
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 представляет собой R, G, S или T
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 представляет собой G, R, L, V или T
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X8 представляет собой T, G, L или отсутствует
<220>
<221> ВАРИАНТ
<222> (9)...(9)
<223> X9 представляет собой A, G, Q или отсутствует
<220>
<221> ВАРИАНТ
<222> (10)...(10)
<223> X10 представляет собой G или отсутствует
<220>
<221> ВАРИАНТ
<222> (11)...(11)
<223> X11 представляет собой G или отсутствует
<220>
<221> ВАРИАНТ
<222> (12)...(12)
<223> X12 представляет собой T или отсутствует
<220>
<221> ВАРИАНТ
<222> (13)...(13)
<223> X13 представляет собой S, T или G
<220>
<221> ВАРИАНТ
<222> (14)...(14)
<223> X14 представляет собой Y, A, G или N
<220>
<221> ВАРИАНТ
<222> (15)...(15)
<223> X15 представляет собой G, S, N, R или E
<220>
<221> ВАРИАНТ
<222> (16)...(16)
<223> X16 представляет собой K или Q
<400> 1206
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5 10 15
Leu Thr
<210> 1207
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR альфа E6(29-38) CDR3 консенсус
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 представляет собой E, T, A, D или L
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 представляет собой G, A, N или R
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 представляет собой R, G, R, T, M или S
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 представляет собой G, V, D, L или отсутствует
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X8 представляет собой T, D или отсутствует
<220>
<221> ВАРИАНТ
<222> (9)...(9)
<223> X9 представляет собой G или отсутствует
<220>
<221> ВАРИАНТ
<222> (10)...(10)
<223> X10 представляет собой S, T, G или отсутствует
<220>
<221> ВАРИАНТ
<222> (11)...(11)
<223> X11 представляет собой G, Y, N, A или W
<220>
<221> ВАРИАНТ
<222> (12)...(12)
<223> X12 представляет собой F, G, N, D, S или Y
<220>
<221> ВАРИАНТ
<222> (13)...(13)
<223> X13 представляет собой K, D,or Q
<220>
<221> ВАРИАНТ
<222> (14)...(14)
<223> X14 представляет собой T, L, M или I
<220>
<221> ВАРИАНТ
<222> (15)...(15)
<223> X15 представляет собой I, T, R или Q
<400> 1207
Ala Met Arg Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5 10 15
<210> 1208
<211> 18
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR альфа E6(29-38) CDR3 консенсус
<220>
<221> ВАРИАНТ
<222> (1)...(1)
<223> X1 представляет собой I или V
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 представляет собой L или V
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 представляет собой L, N или R
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 представляет собой V, F или G
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 представляет собой I, P, G или T
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 представляет собой R, S, P или G
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 представляет собой G, R, Q, T или V
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X8 представляет собой T, G, S или L
<220>
<221> ВАРИАНТ
<222> (9)...(9)
<223> X9 представляет собой A, G, Q или отсутствует
<220>
<221> ВАРИАНТ
<222> (10)...(10)
<223> X10 представляет собой G или отсутствует
<220>
<221> ВАРИАНТ
<222> (11)...(11)
<223> X11 представляет собой G или отсутствует
<220>
<221> ВАРИАНТ
<222> (12)...(12)
<223> X12 представляет собой T или отсутствует
<220>
<221> ВАРИАНТ
<222> (13)...(13)
<223> X13 представляет собой G или S
<220>
<221> ВАРИАНТ
<222> (14)...(14)
<223> X14 представляет собой Y или N
<220>
<221> ВАРИАНТ
<222> (15)...(15)
<223> X15 представляет собой G, Q или E
<220>
<221> ВАРИАНТ
<222> (17)...(17)
<223> X17 представляет собой V или L
<220>
<221> ВАРИАНТ
<222> (18)...(18)
<223> X18 представляет собой I или T
<400> 1208
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Lys
1 5 10 15
Xaa Xaa
<210> 1209
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR альфа E6(29-38) CDR1 консенсус
<220>
<221> ВАРИАНТ
<222> (1)...(1)
<223> X1 представляет собой T, N, D или S
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 представляет собой S, I или R
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 представляет собой D, S, M, A, Y, N или G
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 представляет собой Q, G, P или отсутствует
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 представляет собой S, T, F, I или N
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 представляет собой Y, D, Q, P, N или E
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 представляет собой G, Y, N, S или A
<400> 1209
Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5
<210> 1210
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR альфа E6(29-38) CDR2консенсус
<220>
<221> ВАРИАНТ
<222> (0)...(0)
<220>
<221> ВАРИАНТ
<222> (1)...(1)
<223> X1 представляет собой Q, G, I, V, Y, M, R или N
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 представляет собой G, L, S, Q, Y, T, N или V
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 представляет собой S, T, L или K
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 представляет собой Y, I, S, A, N, F или отсутствует
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 представляет собой D, A или отсутствует
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 представляет собой E, K, Q, S, T, G, D или отсутствует
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 представляет собой Q, S, N, R, G, L или D
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X8 представляет собой N, K, E, V или L
<400> 1210
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5
<210> 1211
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR бета E6(29-38) CDR3 консенсус
<220>
<221> ВАРИАНТ
<222> (1)...(1)
<223> X1 представляет собой A, S или V
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 представляет собой S, A или V
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 представляет собой S, V, R или Q
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 представляет собой H, P, Q, L, Y, G, T, F, S, R или E
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 представляет собой L, G, R, W, F, S, V, T, Y, Q или отсутствует
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 представляет собой A, G, L, T, E, P или отсутствует
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 представляет собой G, T, A, R, Q, N, S или отсутствует
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X8 представляет собой G, S или отсутствует
<220>
<221> ВАРИАНТ
<222> (9)...(9)
<223> X9 представляет собой G или отсутствует
<220>
<221> ВАРИАНТ
<222> (10)...(10)
<223> X10 представляет собой F, G, A, S, T, R, Q, L или отсутствует
<220>
<221> ВАРИАНТ
<222> (11)...(11)
<223> X11 представляет собой T, N, F, A, R, S, G или отсутствует
<220>
<221> ВАРИАНТ
<222> (12)...(12)
<223> X12 представляет собой G, T, L D, Y, N, Q, S или E
<220>
<221> ВАРИАНТ
<222> (13)...(13)
<223> X13 представляет собой E, W, T,G, K, N или P
<220>
<221> ВАРИАНТ
<222> (14)...(14)
<223> X14 представляет собой L, A, K, Q, Y или I
<220>
<221> ВАРИАНТ
<222> (15)...(15)
<223> X15 представляет собой F, H, Y, T или I
<400> 1211
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5 10 15
<210> 1212
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR бета E6(29-38) CDR3 консенсус
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 представляет собой H, P, Q, L, Y, F, R или E
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 представляет собой L, G, R, W, F, S, V, T, Y или Q
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 представляет собой A, G, L, E или P
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 представляет собой G, T, A, R, Q, S или отсутствует
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X8 представляет собой G, S или отсутствует
<220>
<221> ВАРИАНТ
<222> (9)...(9)
<223> X9 представляет собой F, G, A, S, T,R, L или отсутствует
<220>
<221> ВАРИАНТ
<222> (10)...(10)
<223> X10 представляет собой T, N, A, F, R, S или G
<220>
<221> ВАРИАНТ
<222> (11)...(11)
<223> X11 представляет собой G, T, L, D, Y, Q, S, E или N
<220>
<221> ВАРИАНТ
<222> (12)...(12)
<223> X12 представляет собой E, W, T, G, P, или K
<220>
<221> ВАРИАНТ
<222> (13)...(13)
<223> X13 представляет собой L, A, K, Q, Y или I
<220>
<221> ВАРИАНТ
<222> (14)...(14)
<223> X14 представляет собой F, H, Y или T
<400> 1212
Ala Ser Ser Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5 10
<210> 1213
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR бета E6(29-38) CDR3 консенсус
<220>
<221> ВАРИАНТ
<222> (1)...(1)
<223> X1 представляет собой A или S
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 представляет собой S, V или A
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 представляет собой S или V
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 представляет собой L, Y, P или S
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 представляет собой W, F, V, L или Y
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 представляет собой G, T или A
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 представляет собой A, R, Q, S или отсутствует
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X8 представляет собой G или отсутствует
<220>
<221> ВАРИАНТ
<222> (9)...(9)
<223> X9 представляет собой G или отсутствует
<220>
<221> ВАРИАНТ
<222> (10)...(10)
<223> X10 представляет собой S, T, R или G
<220>
<221> ВАРИАНТ
<222> (11)...(11)
<223> X11 представляет собой T, A, R, S или N
<220>
<221> ВАРИАНТ
<222> (12)...(12)
<223> X12 представляет собой D, Y, T или G
<220>
<221> ВАРИАНТ
<222> (13)...(13)
<223> X13 представляет собой T или E
<400> 1213
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Gln Tyr
1 5 10 15
<210> 1214
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR бета E6(29-38) CDR3 консенсус
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 представляет собой S, Q или R
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 представляет собой H, P, T или E
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 представляет собой L, G, W или F
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 представляет собой A, G или отсутствует
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 представляет собой G, N, S, R или отсутствует
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X8 представляет собой F, G, Q, L, A или отсутствует
<220>
<221> ВАРИАНТ
<222> (9)...(9)
<223> X9 представляет собой T, N или A
<220>
<221> ВАРИАНТ
<222> (10)...(10)
<223> X10 представляет собой G, T, N или E
<220>
<221> ВАРИАНТ
<222> (11)...(11)
<223> X11 представляет собой E, N или K
<220>
<221> ВАРИАНТ
<222> (12)...(12)
<223> X12 представляет собой L, A или Q
<400> 1214
Ala Ser Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Phe
1 5 10
<210> 1215
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR бета E6(29-38) CDR3 консенсус
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 представляет собой L или R
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 представляет собой S или T
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 представляет собой G, T или A
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 представляет собой T или отсутствует
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X8 представляет собой G или отсутствует
<220>
<221> ВАРИАНТ
<222> (11)...(11)
<223> X11 представляет собой G или отсутствует
<400> 1215
Ala Ser Ser Xaa Xaa Xaa Xaa Xaa Asn Tyr Xaa Tyr Thr
1 5 10
<210> 1216
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR бета E6(29-38) CDR3 консенсус
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 представляет собой L, F или P
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 представляет собой R или Q
<220>
<221> ВАРИАНТ
<222> (12)...(12)
<223> X12 представляет собой Q или L
<400> 1216
Ala Ser Ser Xaa Trp Gly Xaa Ser Asn Gln Pro Xaa His
1 5 10
<210> 1217
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR бета E6(29-38) CDR3 консенсус
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 представляет собой L или R
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 представляет собой W или Q
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 представляет собой G или P
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 представляет собой R или S
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X8 представляет собой S или отсутствует
<400> 1217
Ala Ser Ser Xaa Xaa Xaa Xaa Xaa Ser Gly Asn Thr Ile Tyr
1 5 10
<210> 1218
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR бета E6(29-38) CDR1 консенсус
<220>
<221> ВАРИАНТ
<222> (1)...(1)
<223> X1 представляет собой S, M, D или L
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 представляет собой G, E, D, N, Q, S или F
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 представляет собой H, V, Y, N или Q
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 представляет собой A, S, F или отсутствует
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 представляет собой W V, N, E, T, P, Y, K, D или L
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 представляет собой S, R, A, N, Y, M или T
<400> 1218
Xaa Xaa Xaa Xaa Xaa Xaa
1 5
<210> 1219
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR бета E6(29-38) CDR2 консенсус
<220>
<221> ВАРИАНТ
<222> (1)...(1)
<223> X1 представляет собой F, Y, S, A или M
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 представляет собой N, Q, V, T, Y или A
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 представляет собой N, D, E, S, G, I, F, Q или L
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 представляет собой G, A, N или отсутствует
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 представляет собой E, K, V, E, S, T, G или N
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 представляет собой A, E, K, G, L, D, V или N
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 представляет собой Q, M, T, A, V, E, P, D или I
<400> 1219
Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5
<210> 1220
<211> 18
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR альфа E6(29-38) CDR3 консенсус
<220>
<221> ВАРИАНТ
<222> (1)...(1)
<223> X1 представляет собой A, I или V
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 представляет собой M, L, A, V, S или E
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 представляет собой R, L, N, S, Q, K, G или W
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 представляет собой E, V, P, T, F, A, G, N, D или L
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 представляет собой G, I, D, L, A, P, N, R, T или отсутствует
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 представляет собой G, N, R, T, M, S, P или отсутствует
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 представляет собой G, V, D, L, Q, T, R или отсутствует
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X8 представляет собой T, D, S, L, G или отсутствует
<220>
<221> ВАРИАНТ
<222> (9)...(9)
<223> X9 представляет собой A, G, Q или отсутствует
<220>
<221> ВАРИАНТ
<222> (10)...(10)
<223> X10 представляет собой G или отсутствует
<220>
<221> ВАРИАНТ
<222> (11)...(11)
<223> X11 представляет собой G или отсутствует
<220>
<221> ВАРИАНТ
<222> (12)...(12)
<223> X12 представляет собой T или отсутствует
<220>
<221> ВАРИАНТ
<222> (13)...(13)
<223> X13 представляет собой S, A, T, G или отсутствует
<220>
<221> ВАРИАНТ
<222> (14)...(14)
<223> X14 представляет собой G, Y, T, N, A, W или отсутствует
<220>
<221> ВАРИАНТ
<222> (15)...(15)
<223> X15 представляет собой F, G, N, T, Y, D, S, R, Q или E
<220>
<221> ВАРИАНТ
<222> (16)...(16)
<223> X16 представляет собой K, P, A, D или Q
<220>
<221> ВАРИАНТ
<222> (17)...(18)
<223> X17 представляет собой L, M, I, V или T
X18 представляет собой I, T, V, F, R или Q
<400> 1220
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5 10 15
Xaa Xaa
<210> 1221
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR альфа E6(296-38) CDR3 консенсус
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 представляет собой V или M
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 представляет собой P или D
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 представляет собой N или R
<400> 1221
Ala Xaa Arg Xaa Ala Xaa Asn Asn Asp Met Arg
1 5 10
<210> 1222
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR альфа E6(29-38) CDR3 консенсус
<220>
<221> ВАРИАНТ
<222> (1)...(1)
<223> X1 представляет собой A, S или V
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 представляет собой S, A или V
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 представляет собой S, R или Q
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 представляет собой H, P, Q, L, Y, G, T, F, S, R или E
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 представляет собой L, G, R, W, F, S, V, T, Y, Q или отсутствует
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 представляет собой A, G, L, E, P или отсутствует
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 представляет собой G, T, A, R, Q, N, S или отсутствует
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X8 представляет собой G, S или отсутствует
<220>
<221> ВАРИАНТ
<222> (9)...(9)
<223> X9 представляет собой G или отсутствует
<220>
<221> ВАРИАНТ
<222> (10)...(10)
<223> X10 представляет собой F, G, A, S, T, R, Q, L или отсутствует
<220>
<221> ВАРИАНТ
<222> (11)...(11)
<223> X11 представляет собой T, N, F, A, R, S, G или отсутствует
<220>
<221> ВАРИАНТ
<222> (12)...(12)
<223> X12 представляет собой G, T, L D, Y, N, Q, S или E
<220>
<221> ВАРИАНТ
<222> (13)...(13)
<223> X13 представляет собой E, W, T,G, K, N или P
<220>
<221> ВАРИАНТ
<222> (14)...(14)
<223> X14 представляет собой L, A, K, Q, Y или I
<220>
<221> ВАРИАНТ
<222> (15)...(15)
<223> X15 представляет собой F, H, Y, T или I
<400> 1222
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5 10 15
<210> 1223
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR бета E6(29-38) CDR3 консенсус
<220>
<221> ВАРИАНТ
<222> (1)...(1)
<223> X1 представляет собой A или S
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 представляет собой S или A
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 представляет собой L, Y, P или S
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 представляет собой W, F, V, L или Y
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 представляет собой G или A
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 представляет собой A, R, Q, S или отсутствует
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X8 представляет собой G или отсутствует
<220>
<221> ВАРИАНТ
<222> (9)...(9)
<223> X9 представляет собой G или отсутствует
<220>
<221> ВАРИАНТ
<222> (10)...(10)
<223> X10 представляет собой S, T, R или G
<220>
<221> ВАРИАНТ
<222> (11)...(11)
<223> X11 представляет собой T, A, R, S или N
<220>
<221> ВАРИАНТ
<222> (12)...(12)
<223> X12 представляет собой D, Y, T или G
<220>
<221> ВАРИАНТ
<222> (13)...(13)
<223> X13 представляет собой T или E
<400> 1223
Xaa Xaa Ser Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Gln Tyr
1 5 10 15
<210> 1224
<211> 450
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 31 - бета нативный
<400> 1224
gacagtgatc ctgatctggt aaagctccca tcctgccctg accctgccat gggcaccagg 60
ctcctcttct gggtggcctt ctgtctcctg ggggcagatc acacaggagc tggagtctcc 120
cagtccccca gtaacaaggt cacagagaag ggaaaggatg tagagctcag gtgtgatcca 180
atttcaggtc atactgccct ttactggtac cgacagagcc tggggcaggg cctggagttt 240
ttaatttact tccaaggcaa cagtgcacca gacaaatcag ggctgcccag tgatcgcttc 300
tctgcagaga ggactggggg atccgtctcc actctgacga tccagcgcac acagcaggag 360
gactcggccg tgtatctctg tgccagcagc cgcttcctcg gaagcacaga tacgcagtat 420
tttggcccag gcacccggct gacagtgctc 450
<210> 1225
<211> 469
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 31 - альфа нативный
<400> 1225
gagatttgca gctttctagg caggagacaa gacaatctgc atcttcacag gagggatggc 60
catgctcctg ggggcatcag tgctgattct gtggcttcag ccagactggg taaacagtca 120
acagaagaat gatgaccagc aagttaagca aaattcacca tccctgagcg tccaggaagg 180
aagaatttct attctgaact gtgactatac taacagcatg tttgattatt tcctatggta 240
caaaaaatac cctgctgaag gtcctacatt cctgatatct ataagttcca ttaaggataa 300
aaatgaagat ggaagattca ctgtcttctt aaacaaaagt gccaagcacc tctctctgca 360
cattgtgccc tcccagcctg gagactctgc agtgtacttc tgtgcagcaa gcgaaagagg 420
aacctacaaa tacatctttg gaacaggcac caggctgaag gttttagca 469
<210> 1226
<211> 449
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 34 - альфа нативный
<400> 1226
gggatagccc tggccaactt tcaaggctcc taaatctgag ttttcagtga actggacaga 60
aaaaaaaaat gaagaagcta ctagcaatga ttctgtggct tcaactagac cggttaagtg 120
gagagctgaa agtggaacaa aaccctctgt tcctgagcat gcaggaggga aaaaactata 180
ccatctactg caattattca accacttcag acagactgta ttggtacagg caggatcctg 240
ggaaaagtct ggaatctctg tttgtgttgc tatcaaatgg agcagtgaag caggagggac 300
gattaatggc ctcacttgat accaaagccc gtctcagcac cctccacatc acagctgccg 360
tgcatgacct ctctgccacc tacttctgtg ccgtgtcagg aacctacaaa tacatctttg 420
gaacaggcac caggctgaag gttttagca 449
<210> 1227
<211> 418
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 34 - бета нативный
<400> 1227
gggctgcctg ccccactgtg ccatgggccc tgggctcctc tgctgggcgc tgctttgtct 60
cctgggagca ggctcagtgg agactggagt cacccaaagt cccacacacc tgatcaaaac 120
gagaggacag caagtgactc tgagatgctc ttctcagtct gggcacaaca ctgtgtcctg 180
gtaccaacag gccctgggtc aggggcccca gtttatcttt cagtattata gggaggaaga 240
gaatggcagg ggaaacttcc ctcctagatt ctcaggtctc cagttcccta attatagctc 300
tgagctgaat gtgaacgcct tggagctgga cgactcggcc ctgtatctct gtgccagcag 360
ctcgtatgcc ggaagctacg agcagtactt cgggccgggc accaggctca cggtcaca 418
<210> 1228
<211> 432
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 55 - бета нативный
<400> 1228
gattcctgct gtgatcctgc catggatacc tggctcgtat gctgggcaat ttttagtctc 60
ttgaaagcag gactcacaga acctgaagtc acccagactc ccagccatca ggtcacacag 120
atgggacagg aagtgatctt gcgctgtgtc cccatctcta atcacttata cttctattgg 180
tacagacaaa tcttggggca gaaagtcgag tttctggttt ccttttataa taatgaaatc 240
tcagagaagt ctgaaatatt cgatgatcaa ttctcagttg aaaggcctga tggatcaaat 300
ttcactctga agatccggtc cacaaagctg gaggactcag ccatgtactt ctgtgccagc 360
acaacgagga gctcctacga gcagtacttc gggccgggca ccaggctcac ggtcacagag 420
gacctgaaaa ac 432
<210> 1229
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRAC-10
<400> 1229
ucucucagcu gguacacggc 20
<210> 1230
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRAC-110
<400> 1230
uggauuuaga gucucucagc 20
<210> 1231
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRAC-116
<400> 1231
acacggcagg gucaggguuc 20
<210> 1232
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRAC-4
<400> 1232
gcugguacac ggcaggguca 20
<210> 1233
<211> 17
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRAC-49
<400> 1233
cucagcuggu acacggc 17
<210> 1234
<211> 17
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRAC-2
<400> 1234
ugguacacgg caggguc 17
<210> 1235
<211> 17
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRAC-30
<400> 1235
gcuagacaug aggucua 17
<210> 1236
<211> 17
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRAC-43
<400> 1236
gucagauuug uugcucc 17
<210> 1237
<211> 17
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRAC-23
<400> 1237
ucagcuggua cacggca 17
<210> 1238
<211> 17
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRAC-34
<400> 1238
gcagacagac uugucac 17
<210> 1239
<211> 17
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRAC-25
<400> 1239
gguacacggc aggguca 17
<210> 1240
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRAC-128
<400> 1240
cuucaagagc aacagugcug 20
<210> 1241
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRAC-105
<400> 1241
agagcaacag ugcuguggcc 20
<210> 1242
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRAC-106
<400> 1242
aaagucagau uuguugcucc 20
<210> 1243
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRAC-123
<400> 1243
acaaaacugu gcuagacaug 20
<210> 1244
<211> 17
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRAC-64
<400> 1244
aaacugugcu agacaug 17
<210> 1245
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRAC-97
<400> 1245
ugugcuagac augaggucua 20
<210> 1246
<211> 22
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRAC-148
<400> 1246
ggcuggggaa gaaggugucu uc 22
<210> 1247
<211> 21
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRAC-147
<400> 1247
gcuggggaag aaggugucuu c 21
<210> 1248
<211> 18
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRAC-234
<400> 1248
ggggaagaag gugucuuc 18
<210> 1249
<211> 22
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRAC-167
<400> 1249
guuuugucug ugauauacac au 22
<210> 1250
<211> 24
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRAC-177
<400> 1250
ggcagacaga cuugucacug gauu 24
<210> 1251
<211> 23
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRAC-176
<400> 1251
gcagacagac uugucacugg auu 23
<210> 1252
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRAC-257
<400> 1252
gacagacuug ucacuggauu 20
<210> 1253
<211> 24
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRAC-233
<400> 1253
gugaauaggc agacagacuu guca 24
<210> 1254
<211> 22
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRAC-231
<400> 1254
gaauaggcag acagacuugu ca 22
<210> 1255
<211> 22
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRAC-163
<400> 1255
gagucucuca gcugguacac gg 22
<210> 1256
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRAC-241
<400> 1256
gucucucagc ugguacacgg 20
<210> 1257
<211> 22
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRAC-179
<400> 1257
gguacacggc agggucaggg uu 22
<210> 1258
<211> 21
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRAC-178
<400> 1258
guacacggca gggucagggu u 21
<210> 1259
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-40
<400> 1259
cacccagauc gucagcgccg 20
<210> 1260
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-52
<400> 1260
caaacacagc gaccucgggu 20
<210> 1261
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-25
<400> 1261
ugacgagugg acccaggaua 20
<210> 1262
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-35
<400> 1262
ggcucucgga gaaugacgag 20
<210> 1263
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-39
<400> 1263
gaaaaacgug uucccacccg 20
<210> 1264
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-49
<400> 1264
augacgagug gacccaggau 20
<210> 1265
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-51
<400> 1265
aguccaguuc uacgggcucu 20
<210> 1266
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-26
<400> 1266
cgcugucaag uccaguucua 20
<210> 1267
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-47
<400> 1267
aucgucagcg ccgaggccug 20
<210> 1268
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-45
<400> 1268
ucaaacacag cgaccucggg 20
<210> 1269
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-34
<400> 1269
cguagaacug gacuugacag 20
<210> 1270
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-227
<400> 1270
aggccucggc gcugacgauc 20
<210> 1271
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-41
<400> 1271
ugacagcgga agugguugcg 20
<210> 1272
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-30
<400> 1272
uugacagcgg aagugguugc 20
<210> 1273
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-206
<400> 1273
ucuccgagag cccguagaac 20
<210> 1274
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-32
<400> 1274
cgggugggaa cacguuuuuc 20
<210> 1275
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-276
<400> 1275
gacagguuug gcccuauccu 20
<210> 1276
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-274
<400> 1276
gaucgucagc gccgaggccu 20
<210> 1277
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-230
<400> 1277
ggcucaaaca cagcgaccuc 20
<210> 1278
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-235
<400> 1278
ugagggucuc ggccaccuuc 20
<210> 1279
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-38
<400> 1279
aggcuucuac cccgaccacg 20
<210> 1280
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-223
<400> 1280
ccgaccacgu ggagcugagc 20
<210> 1281
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-221
<400> 1281
ugacagguuu ggcccuaucc 20
<210> 1282
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-48
<400> 1282
cuugacagcg gaagugguug 20
<210> 1283
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-216
<400> 1283
agaucgucag cgccgaggcc 20
<210> 1284
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-210
<400> 1284
gcgcugacga ucugggugac 20
<210> 1285
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-268
<400> 1285
ugagggcggg cugcuccuug 20
<210> 1286
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-193
<400> 1286
guugcggggg uucugccaga 20
<210> 1287
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-246
<400> 1287
agcucagcuc cacguggucg 20
<210> 1288
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-228
<400> 1288
gcggcugcuc aggcaguauc 20
<210> 1289
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-43
<400> 1289
gcggggguuc ugccagaagg 20
<210> 1290
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-272
<400> 1290
uggcucaaac acagcgaccu 20
<210> 1291
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-33
<400> 1291
acuggacuug acagcggaag 20
<210> 1292
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-44
<400> 1292
gacagcggaa gugguugcgg 20
<210> 1293
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-211
<400> 1293
gcugucaagu ccaguucuac 20
<210> 1294
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-253
<400> 1294
guaucuggag ucauugaggg 20
<210> 1295
<211> 17
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-18
<400> 1295
cucggcgcug acgaucu 17
<210> 1296
<211> 17
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-6
<400> 1296
ccucggcgcu gacgauc 17
<210> 1297
<211> 17
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-85
<400> 1297
ccgagagccc guagaac 17
<210> 1298
<211> 17
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-129
<400> 1298
ccagaucguc agcgccg 17
<210> 1299
<211> 17
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-93
<400> 1299
gaaugacgag uggaccc 17
<210> 1300
<211> 22
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-415
<400> 1300
gggugacagg uuuggcccua uc 22
<210> 1301
<211> 21
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-414
<400> 1301
ggugacaggu uuggcccuau c 21
<210> 1302
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-310
<400> 1302
gugacagguu uggcccuauc 20
<210> 1303
<211> 18
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-308
<400> 1303
gacagguuug gcccuauc 18
<210> 1304
<211> 22
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-401
<400> 1304
gauacugccu gagcagccgc cu 22
<210> 1305
<211> 24
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-468
<400> 1305
gaccacgugg agcugagcug gugg 24
<210> 1306
<211> 18
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-462
<400> 1306
guggagcuga gcuggugg 18
<210> 1307
<211> 24
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-424
<400> 1307
gggcgggcug cuccuugagg ggcu 24
<210> 1308
<211> 23
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-423
<400> 1308
ggcgggcugc uccuugaggg gcu 23
<210> 1309
<211> 22
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-422
<400> 1309
gcgggcugcu ccuugagggg cu 22
<210> 1310
<211> 20
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-420
<400> 1310
gggcugcucc uugaggggcu 20
<210> 1311
<211> 19
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-419
<400> 1311
ggcugcuccu ugaggggcu 19
<210> 1312
<211> 18
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-418
<400> 1312
gcugcuccuu gaggggcu 18
<210> 1313
<211> 24
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-445
<400> 1313
ggugaauggg aaggaggugc acag 24
<210> 1314
<211> 23
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-444
<400> 1314
gugaauggga aggaggugca cag 23
<210> 1315
<211> 21
<212> РНК
<213> Искусственная последовательность
<220>
<223> TRBC-442
<400> 1315
gaaugggaag gaggugcaca g 21
<210> 1316
<211> 96
<212> РНК
<213> Искусственная последовательность
<220>
<223> иллюстративная гРНК
<220>
<221> прочие_признаки
<222> (1)...(20)
<223> n представляет собой a, c, u, g, неизвестно или другое
<400> 1316
nnnnnnnnnn nnnnnnnnnn guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1317
<211> 104
<212> РНК
<213> Искусственная последовательность
<220>
<223> иллюстративная гРНК
<220>
<221> прочие_признаки
<222> (1)...(20)
<223> n представляет собой a, c, u, g, неизвестно или другое
<400> 1317
nnnnnnnnnn nnnnnnnnnn guuuuagagc uaugcugaaa agcauagcaa guuaaaauaa 60
ggcuaguccg uuaucaacuu gaaaaagugg caccgagucg gugc 104
<210> 1318
<211> 106
<212> РНК
<213> Искусственная последовательность
<220>
<223> иллюстративная гРНК
<220>
<221> прочие_признаки
<222> (1)...(20)
<223> n представляет собой a, c, u, g, неизвестно или другое
<400> 1318
nnnnnnnnnn nnnnnnnnnn guuuuagagc uaugcuggaa acagcauagc aaguuaaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1319
<211> 116
<212> РНК
<213> Искусственная последовательность
<220>
<223> иллюстративная гРНК
<220>
<221> прочие_признаки
<222> (1)...(20)
<223> n представляет собой a, c, u, g, неизвестно или другое
<400> 1319
nnnnnnnnnn nnnnnnnnnn guuuuagagc uaugcuguuu uggaaacaaa acagcauagc 60
aaguuaaaau aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 116
<210> 1320
<211> 96
<212> РНК
<213> Искусственная последовательность
<220>
<223> иллюстративная гРНК
<220>
<221> прочие_признаки
<222> (1)...(20)
<223> n представляет собой a, c, u, g, неизвестно или другое
<400> 1320
nnnnnnnnnn nnnnnnnnnn guauuagagc uagaaauagc aaguuaauau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1321
<211> 96
<212> РНК
<213> Искусственная последовательность
<220>
<223> иллюстративная гРНК
<220>
<221> прочие_признаки
<222> (1)...(20)
<223> n представляет собой a, c, u, g, неизвестно или другое
<400> 1321
nnnnnnnnnn nnnnnnnnnn guuuaagagc uagaaauagc aaguuuaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1322
<211> 116
<212> РНК
<213> Искусственная последовательность
<220>
<223> иллюстративная гРНК
<220>
<221> прочие_признаки
<222> (1)...(20)
<223> n представляет собой a, c, u, g, неизвестно или другое
<400> 1322
nnnnnnnnnn nnnnnnnnnn guauuagagc uaugcuguau uggaaacaau acagcauagc 60
aaguuaauau aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 116
<210> 1323
<211> 47
<212> РНК
<213> Искусственная последовательность
<220>
<223> иллюстративные проксимальные и хвостовые домены
<400> 1323
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugcu 47
<210> 1324
<211> 49
<212> РНК
<213> Искусственная последовательность
<220>
<223> иллюстративные проксимальные и хвостовые домены
<400> 1324
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cgguggugc 49
<210> 1325
<211> 51
<212> РНК
<213> Искусственная последовательность
<220>
<223> иллюстративные проксимальные и хвостовые домены
<400> 1325
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugcggau c 51
<210> 1326
<211> 31
<212> РНК
<213> Искусственная последовательность
<220>
<223> иллюстративные проксимальные и хвостовые домены
<400> 1326
aaggcuaguc cguuaucaac uugaaaaagu g 31
<210> 1327
<211> 18
<212> РНК
<213> Искусственная последовательность
<220>
<223> иллюстративные проксимальные и хвостовые домены
<400> 1327
aaggcuaguc cguuauca 18
<210> 1328
<211> 12
<212> РНК
<213> Искусственная последовательность
<220>
<223> иллюстративные проксимальные и хвостовые домены
<400> 1328
aaggcuaguc cg 12
<210> 1329
<211> 102
<212> РНК
<213> Искусственная последовательность
<220>
<223> иллюстративная гРНК
<220>
<221> прочие_признаки
<222> (1)...(20)
<223> n представляет собой a, c, u, g, неизвестно или другое
<400> 1329
nnnnnnnnnn nnnnnnnnnn guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcuuuu uu 102
<210> 1330
<211> 102
<212> РНК
<213> Искусственная последовательность
<220>
<223> иллюстративная гРНК
<220>
<221> прочие_связывания
<222> (1)...(20)
<223> n представляет собой a, c, u, g, неизвестно или другое
<400> 1330
nnnnnnnnnn nnnnnnnnnn guuuuaguac ucuggaaaca gaaucuacua aaacaaggca 60
aaaugccgug uuuaucucgu caacuuguug gcgagauuuu uu 102
<210> 1331
<211> 1344
<212> Белок
<213> Streptococcus mutans
<400> 1331
Lys Lys Pro Tyr Ser Ile Gly Leu Asp Ile Gly Thr Asn Ser Val Gly
1 5 10 15
Trp Ala Val Val Thr Asp Asp Tyr Lys Val Pro Ala Lys Lys Met Lys
20 25 30
Val Leu Gly Asn Thr Asp Lys Ser His Ile Glu Lys Asn Leu Leu Gly
35 40 45
Ala Leu Leu Phe Asp Ser Gly Asn Thr Ala Glu Asp Arg Arg Leu Lys
50 55 60
Arg Thr Ala Arg Arg Arg Tyr Thr Arg Arg Arg Asn Arg Ile Leu Tyr
65 70 75 80
Leu Gln Glu Ile Phe Ser Glu Glu Met Gly Lys Val Asp Asp Ser Phe
85 90 95
Phe His Arg Leu Glu Asp Ser Phe Leu Val Thr Glu Asp Lys Arg Gly
100 105 110
Glu Arg His Pro Ile Phe Gly Asn Leu Glu Glu Glu Val Lys Tyr His
115 120 125
Glu Asn Phe Pro Thr Ile Tyr His Leu Arg Gln Tyr Leu Ala Asp Asn
130 135 140
Pro Glu Lys Val Asp Leu Arg Leu Val Tyr Leu Ala Leu Ala His Ile
145 150 155 160
Ile Lys Phe Arg Gly His Phe Leu Ile Glu Gly Lys Phe Asp Thr Arg
165 170 175
Asn Asn Asp Val Gln Arg Leu Phe Gln Glu Phe Leu Ala Val Tyr Asp
180 185 190
Asn Thr Phe Glu Asn Ser Ser Leu Gln Glu Gln Asn Val Gln Val Glu
195 200 205
Glu Ile Leu Thr Asp Lys Ile Ser Lys Ser Ala Lys Lys Asp Arg Val
210 215 220
Leu Lys Leu Phe Pro Asn Glu Lys Ser Asn Gly Arg Phe Ala Glu Phe
225 230 235 240
Leu Lys Leu Ile Val Gly Asn Gln Ala Asp Phe Lys Lys His Phe Glu
245 250 255
Leu Glu Glu Lys Ala Pro Leu Gln Phe Ser Lys Asp Thr Tyr Glu Glu
260 265 270
Glu Leu Glu Val Leu Leu Ala Gln Ile Gly Asp Asn Tyr Ala Glu Leu
275 280 285
Phe Leu Ser Ala Lys Lys Leu Tyr Asp Ser Ile Leu Leu Ser Gly Ile
290 295 300
Leu Thr Val Thr Asp Val Gly Thr Lys Ala Pro Leu Ser Ala Ser Met
305 310 315 320
Ile Gln Arg Tyr Asn Glu His Gln Met Asp Leu Ala Gln Leu Lys Gln
325 330 335
Phe Ile Arg Gln Lys Leu Ser Asp Lys Tyr Asn Glu Val Phe Ser Asp
340 345 350
Val Ser Lys Asp Gly Tyr Ala Gly Tyr Ile Asp Gly Lys Thr Asn Gln
355 360 365
Glu Ala Phe Tyr Lys Tyr Leu Lys Gly Leu Leu Asn Lys Ile Glu Gly
370 375 380
Ser Gly Tyr Phe Leu Asp Lys Ile Glu Arg Glu Asp Phe Leu Arg Lys
385 390 395 400
Gln Arg Thr Phe Asp Asn Gly Ser Ile Pro His Gln Ile His Leu Gln
405 410 415
Glu Met Arg Ala Ile Ile Arg Arg Gln Ala Glu Phe Tyr Pro Phe Leu
420 425 430
Ala Asp Asn Gln Asp Arg Ile Glu Lys Leu Leu Thr Phe Arg Ile Pro
435 440 445
Tyr Tyr Val Gly Pro Leu Ala Arg Gly Lys Ser Asp Phe Ala Trp Leu
450 455 460
Ser Arg Lys Ser Ala Asp Lys Ile Thr Pro Trp Asn Phe Asp Glu Ile
465 470 475 480
Val Asp Lys Glu Ser Ser Ala Glu Ala Phe Ile Asn Arg Met Thr Asn
485 490 495
Tyr Asp Leu Tyr Leu Pro Asn Gln Lys Val Leu Pro Lys His Ser Leu
500 505 510
Leu Tyr Glu Lys Phe Thr Val Tyr Asn Glu Leu Thr Lys Val Lys Tyr
515 520 525
Lys Thr Glu Gln Gly Lys Thr Ala Phe Phe Asp Ala Asn Met Lys Gln
530 535 540
Glu Ile Phe Asp Gly Val Phe Lys Val Tyr Arg Lys Val Thr Lys Asp
545 550 555 560
Lys Leu Met Asp Phe Leu Glu Lys Glu Phe Asp Glu Phe Arg Ile Val
565 570 575
Asp Leu Thr Gly Leu Asp Lys Glu Asn Lys Val Phe Asn Ala Ser Tyr
580 585 590
Gly Thr Tyr His Asp Leu Cys Lys Ile Leu Asp Lys Asp Phe Leu Asp
595 600 605
Asn Ser Lys Asn Glu Lys Ile Leu Glu Asp Ile Val Leu Thr Leu Thr
610 615 620
Leu Phe Glu Asp Arg Glu Met Ile Arg Lys Arg Leu Glu Asn Tyr Ser
625 630 635 640
Asp Leu Leu Thr Lys Glu Gln Val Lys Lys Leu Glu Arg Arg His Tyr
645 650 655
Thr Gly Trp Gly Arg Leu Ser Ala Glu Leu Ile His Gly Ile Arg Asn
660 665 670
Lys Glu Ser Arg Lys Thr Ile Leu Asp Tyr Leu Ile Asp Asp Gly Asn
675 680 685
Ser Asn Arg Asn Phe Met Gln Leu Ile Asn Asp Asp Ala Leu Ser Phe
690 695 700
Lys Glu Glu Ile Ala Lys Ala Gln Val Ile Gly Glu Thr Asp Asn Leu
705 710 715 720
Asn Gln Val Val Ser Asp Ile Ala Gly Ser Pro Ala Ile Lys Lys Gly
725 730 735
Ile Leu Gln Ser Leu Lys Ile Val Asp Glu Leu Val Lys Ile Met Gly
740 745 750
His Gln Pro Glu Asn Ile Val Val Glu Met Ala Arg Glu Asn Gln Phe
755 760 765
Thr Asn Gln Gly Arg Arg Asn Ser Gln Gln Arg Leu Lys Gly Leu Thr
770 775 780
Asp Ser Ile Lys Glu Phe Gly Ser Gln Ile Leu Lys Glu His Pro Val
785 790 795 800
Glu Asn Ser Gln Leu Gln Asn Asp Arg Leu Phe Leu Tyr Tyr Leu Gln
805 810 815
Asn Gly Arg Asp Met Tyr Thr Gly Glu Glu Leu Asp Ile Asp Tyr Leu
820 825 830
Ser Gln Tyr Asp Ile Asp His Ile Ile Pro Gln Ala Phe Ile Lys Asp
835 840 845
Asn Ser Ile Asp Asn Arg Val Leu Thr Ser Ser Lys Glu Asn Arg Gly
850 855 860
Lys Ser Asp Asp Val Pro Ser Lys Asp Val Val Arg Lys Met Lys Ser
865 870 875 880
Tyr Trp Ser Lys Leu Leu Ser Ala Lys Leu Ile Thr Gln Arg Lys Phe
885 890 895
Asp Asn Leu Thr Lys Ala Glu Arg Gly Gly Leu Thr Asp Asp Asp Lys
900 905 910
Ala Gly Phe Ile Lys Arg Gln Leu Val Glu Thr Arg Gln Ile Thr Lys
915 920 925
His Val Ala Arg Ile Leu Asp Glu Arg Phe Asn Thr Glu Thr Asp Glu
930 935 940
Asn Asn Lys Lys Ile Arg Gln Val Lys Ile Val Thr Leu Lys Ser Asn
945 950 955 960
Leu Val Ser Asn Phe Arg Lys Glu Phe Glu Leu Tyr Lys Val Arg Glu
965 970 975
Ile Asn Asp Tyr His His Ala His Asp Ala Tyr Leu Asn Ala Val Ile
980 985 990
Gly Lys Ala Leu Leu Gly Val Tyr Pro Gln Leu Glu Pro Glu Phe Val
995 1000 1005
Tyr Gly Asp Tyr Pro His Phe His Gly His Lys Glu Asn Lys Ala Thr
1010 1015 1020
Ala Lys Lys Phe Phe Tyr Ser Asn Ile Met Asn Phe Phe Lys Lys Asp
1025 1030 1035 1040
Asp Val Arg Thr Asp Lys Asn Gly Glu Ile Ile Trp Lys Lys Asp Glu
1045 1050 1055
His Ile Ser Asn Ile Lys Lys Val Leu Ser Tyr Pro Gln Val Asn Ile
1060 1065 1070
Val Lys Lys Val Glu Glu Gln Thr Gly Gly Phe Ser Lys Glu Ser Ile
1075 1080 1085
Leu Pro Lys Gly Asn Ser Asp Lys Leu Ile Pro Arg Lys Thr Lys Lys
1090 1095 1100
Phe Tyr Trp Asp Thr Lys Lys Tyr Gly Gly Phe Asp Ser Pro Ile Val
1105 1110 1115 1120
Ala Tyr Ser Ile Leu Val Ile Ala Asp Ile Glu Lys Gly Lys Ser Lys
1125 1130 1135
Lys Leu Lys Thr Val Lys Ala Leu Val Gly Val Thr Ile Met Glu Lys
1140 1145 1150
Met Thr Phe Glu Arg Asp Pro Val Ala Phe Leu Glu Arg Lys Gly Tyr
1155 1160 1165
Arg Asn Val Gln Glu Glu Asn Ile Ile Lys Leu Pro Lys Tyr Ser Leu
1170 1175 1180
Phe Lys Leu Glu Asn Gly Arg Lys Arg Leu Leu Ala Ser Ala Arg Glu
1185 1190 1195 1200
Leu Gln Lys Gly Asn Glu Ile Val Leu Pro Asn His Leu Gly Thr Leu
1205 1210 1215
Leu Tyr His Ala Lys Asn Ile His Lys Val Asp Glu Pro Lys His Leu
1220 1225 1230
Asp Tyr Val Asp Lys His Lys Asp Glu Phe Lys Glu Leu Leu Asp Val
1235 1240 1245
Val Ser Asn Phe Ser Lys Lys Tyr Thr Leu Ala Glu Gly Asn Leu Glu
1250 1255 1260
Lys Ile Lys Glu Leu Tyr Ala Gln Asn Asn Gly Glu Asp Leu Lys Glu
1265 1270 1275 1280
Leu Ala Ser Ser Phe Ile Asn Leu Leu Thr Phe Thr Ala Ile Gly Ala
1285 1290 1295
Pro Ala Thr Phe Lys Phe Phe Asp Lys Asn Ile Asp Arg Lys Arg Tyr
1300 1305 1310
Thr Ser Thr Thr Glu Ile Leu Asn Ala Thr Leu Ile His Gln Ser Ile
1315 1320 1325
Thr Gly Leu Tyr Glu Thr Arg Ile Asp Leu Asn Lys Leu Gly Gly Asp
1330 1335 1340
<210> 1332
<211> 1367
<212> Белок
<213> Streptococcus pyogenes
<400> 1332
Asp Lys Lys Tyr Ser Ile Gly Leu Asp Ile Gly Thr Asn Ser Val Gly
1 5 10 15
Trp Ala Val Ile Thr Asp Glu Tyr Lys Val Pro Ser Lys Lys Phe Lys
20 25 30
Val Leu Gly Asn Thr Asp Arg His Ser Ile Lys Lys Asn Leu Ile Gly
35 40 45
Ala Leu Leu Phe Asp Ser Gly Glu Thr Ala Glu Ala Thr Arg Leu Lys
50 55 60
Arg Thr Ala Arg Arg Arg Tyr Thr Arg Arg Lys Asn Arg Ile Cys Tyr
65 70 75 80
Leu Gln Glu Ile Phe Ser Asn Glu Met Ala Lys Val Asp Asp Ser Phe
85 90 95
Phe His Arg Leu Glu Glu Ser Phe Leu Val Glu Glu Asp Lys Lys His
100 105 110
Glu Arg His Pro Ile Phe Gly Asn Ile Val Asp Glu Val Ala Tyr His
115 120 125
Glu Lys Tyr Pro Thr Ile Tyr His Leu Arg Lys Lys Leu Val Asp Ser
130 135 140
Thr Asp Lys Ala Asp Leu Arg Leu Ile Tyr Leu Ala Leu Ala His Met
145 150 155 160
Ile Lys Phe Arg Gly His Phe Leu Ile Glu Gly Asp Leu Asn Pro Asp
165 170 175
Asn Ser Asp Val Asp Lys Leu Phe Ile Gln Leu Val Gln Thr Tyr Asn
180 185 190
Gln Leu Phe Glu Glu Asn Pro Ile Asn Ala Ser Gly Val Asp Ala Lys
195 200 205
Ala Ile Leu Ser Ala Arg Leu Ser Lys Ser Arg Arg Leu Glu Asn Leu
210 215 220
Ile Ala Gln Leu Pro Gly Glu Lys Lys Asn Gly Leu Phe Gly Asn Leu
225 230 235 240
Ile Ala Leu Ser Leu Gly Leu Thr Pro Asn Phe Lys Ser Asn Phe Asp
245 250 255
Leu Ala Glu Asp Ala Lys Leu Gln Leu Ser Lys Asp Thr Tyr Asp Asp
260 265 270
Asp Leu Asp Asn Leu Leu Ala Gln Ile Gly Asp Gln Tyr Ala Asp Leu
275 280 285
Phe Leu Ala Ala Lys Asn Leu Ser Asp Ala Ile Leu Leu Ser Asp Ile
290 295 300
Leu Arg Val Asn Thr Glu Ile Thr Lys Ala Pro Leu Ser Ala Ser Met
305 310 315 320
Ile Lys Arg Tyr Asp Glu His His Gln Asp Leu Thr Leu Leu Lys Ala
325 330 335
Leu Val Arg Gln Gln Leu Pro Glu Lys Tyr Lys Glu Ile Phe Phe Asp
340 345 350
Gln Ser Lys Asn Gly Tyr Ala Gly Tyr Ile Asp Gly Gly Ala Ser Gln
355 360 365
Glu Glu Phe Tyr Lys Phe Ile Lys Pro Ile Leu Glu Lys Met Asp Gly
370 375 380
Thr Glu Glu Leu Leu Val Lys Leu Asn Arg Glu Asp Leu Leu Arg Lys
385 390 395 400
Gln Arg Thr Phe Asp Asn Gly Ser Ile Pro His Gln Ile His Leu Gly
405 410 415
Glu Leu His Ala Ile Leu Arg Arg Gln Glu Asp Phe Tyr Pro Phe Leu
420 425 430
Lys Asp Asn Arg Glu Lys Ile Glu Lys Ile Leu Thr Phe Arg Ile Pro
435 440 445
Tyr Tyr Val Gly Pro Leu Ala Arg Gly Asn Ser Arg Phe Ala Trp Met
450 455 460
Thr Arg Lys Ser Glu Glu Thr Ile Thr Pro Trp Asn Phe Glu Glu Val
465 470 475 480
Val Asp Lys Gly Ala Ser Ala Gln Ser Phe Ile Glu Arg Met Thr Asn
485 490 495
Phe Asp Lys Asn Leu Pro Asn Glu Lys Val Leu Pro Lys His Ser Leu
500 505 510
Leu Tyr Glu Tyr Phe Thr Val Tyr Asn Glu Leu Thr Lys Val Lys Tyr
515 520 525
Val Thr Glu Gly Met Arg Lys Pro Ala Phe Leu Ser Gly Glu Gln Lys
530 535 540
Lys Ala Ile Val Asp Leu Leu Phe Lys Thr Asn Arg Lys Val Thr Val
545 550 555 560
Lys Gln Leu Lys Glu Asp Tyr Phe Lys Lys Ile Glu Cys Phe Asp Ser
565 570 575
Val Glu Ile Ser Gly Val Glu Asp Arg Phe Asn Ala Ser Leu Gly Thr
580 585 590
Tyr His Asp Leu Leu Lys Ile Ile Lys Asp Lys Asp Phe Leu Asp Asn
595 600 605
Glu Glu Asn Glu Asp Ile Leu Glu Asp Ile Val Leu Thr Leu Thr Leu
610 615 620
Phe Glu Asp Arg Glu Met Ile Glu Glu Arg Leu Lys Thr Tyr Ala His
625 630 635 640
Leu Phe Asp Asp Lys Val Met Lys Gln Leu Lys Arg Arg Arg Tyr Thr
645 650 655
Gly Trp Gly Arg Leu Ser Arg Lys Leu Ile Asn Gly Ile Arg Asp Lys
660 665 670
Gln Ser Gly Lys Thr Ile Leu Asp Phe Leu Lys Ser Asp Gly Phe Ala
675 680 685
Asn Arg Asn Phe Met Gln Leu Ile His Asp Asp Ser Leu Thr Phe Lys
690 695 700
Glu Asp Ile Gln Lys Ala Gln Val Ser Gly Gln Gly Asp Ser Leu His
705 710 715 720
Glu His Ile Ala Asn Leu Ala Gly Ser Pro Ala Ile Lys Lys Gly Ile
725 730 735
Leu Gln Thr Val Lys Val Val Asp Glu Leu Val Lys Val Met Gly Arg
740 745 750
His Lys Pro Glu Asn Ile Val Ile Glu Met Ala Arg Glu Asn Gln Thr
755 760 765
Thr Gln Lys Gly Gln Lys Asn Ser Arg Glu Arg Met Lys Arg Ile Glu
770 775 780
Glu Gly Ile Lys Glu Leu Gly Ser Gln Ile Leu Lys Glu His Pro Val
785 790 795 800
Glu Asn Thr Gln Leu Gln Asn Glu Lys Leu Tyr Leu Tyr Tyr Leu Gln
805 810 815
Asn Gly Arg Asp Met Tyr Val Asp Gln Glu Leu Asp Ile Asn Arg Leu
820 825 830
Ser Asp Tyr Asp Val Asp His Ile Val Pro Gln Ser Phe Leu Lys Asp
835 840 845
Asp Ser Ile Asp Asn Lys Val Leu Thr Arg Ser Asp Lys Asn Arg Gly
850 855 860
Lys Ser Asp Asn Val Pro Ser Glu Glu Val Val Lys Lys Met Lys Asn
865 870 875 880
Tyr Trp Arg Gln Leu Leu Asn Ala Lys Leu Ile Thr Gln Arg Lys Phe
885 890 895
Asp Asn Leu Thr Lys Ala Glu Arg Gly Gly Leu Ser Glu Leu Asp Lys
900 905 910
Ala Gly Phe Ile Lys Arg Gln Leu Val Glu Thr Arg Gln Ile Thr Lys
915 920 925
His Val Ala Gln Ile Leu Asp Ser Arg Met Asn Thr Lys Tyr Asp Glu
930 935 940
Asn Asp Lys Leu Ile Arg Glu Val Lys Val Ile Thr Leu Lys Ser Lys
945 950 955 960
Leu Val Ser Asp Phe Arg Lys Asp Phe Gln Phe Tyr Lys Val Arg Glu
965 970 975
Ile Asn Asn Tyr His His Ala His Asp Ala Tyr Leu Asn Ala Val Val
980 985 990
Gly Thr Ala Leu Ile Lys Lys Tyr Pro Lys Leu Glu Ser Glu Phe Val
995 1000 1005
Tyr Gly Asp Tyr Lys Val Tyr Asp Val Arg Lys Met Ile Ala Lys Ser
1010 1015 1020
Glu Gln Glu Ile Gly Lys Ala Thr Ala Lys Tyr Phe Phe Tyr Ser Asn
1025 1030 1035 1040
Ile Met Asn Phe Phe Lys Thr Glu Ile Thr Leu Ala Asn Gly Glu Ile
1045 1050 1055
Arg Lys Arg Pro Leu Ile Glu Thr Asn Gly Glu Thr Gly Glu Ile Val
1060 1065 1070
Trp Asp Lys Gly Arg Asp Phe Ala Thr Val Arg Lys Val Leu Ser Met
1075 1080 1085
Pro Gln Val Asn Ile Val Lys Lys Thr Glu Val Gln Thr Gly Gly Phe
1090 1095 1100
Ser Lys Glu Ser Ile Leu Pro Lys Arg Asn Ser Asp Lys Leu Ile Ala
1105 1110 1115 1120
Arg Lys Lys Asp Trp Asp Pro Lys Lys Tyr Gly Gly Phe Asp Ser Pro
1125 1130 1135
Thr Val Ala Tyr Ser Val Leu Val Val Ala Lys Val Glu Lys Gly Lys
1140 1145 1150
Ser Lys Lys Leu Lys Ser Val Lys Glu Leu Leu Gly Ile Thr Ile Met
1155 1160 1165
Glu Arg Ser Ser Phe Glu Lys Asn Pro Ile Asp Phe Leu Glu Ala Lys
1170 1175 1180
Gly Tyr Lys Glu Val Lys Lys Asp Leu Ile Ile Lys Leu Pro Lys Tyr
1185 1190 1195 1200
Ser Leu Phe Glu Leu Glu Asn Gly Arg Lys Arg Met Leu Ala Ser Ala
1205 1210 1215
Gly Glu Leu Gln Lys Gly Asn Glu Leu Ala Leu Pro Ser Lys Tyr Val
1220 1225 1230
Asn Phe Leu Tyr Leu Ala Ser His Tyr Glu Lys Leu Lys Gly Ser Pro
1235 1240 1245
Glu Asp Asn Glu Gln Lys Gln Leu Phe Val Glu Gln His Lys His Tyr
1250 1255 1260
Leu Asp Glu Ile Ile Glu Gln Ile Ser Glu Phe Ser Lys Arg Val Ile
1265 1270 1275 1280
Leu Ala Asp Ala Asn Leu Asp Lys Val Leu Ser Ala Tyr Asn Lys His
1285 1290 1295
Arg Asp Lys Pro Ile Arg Glu Gln Ala Glu Asn Ile Ile His Leu Phe
1300 1305 1310
Thr Leu Thr Asn Leu Gly Ala Pro Ala Ala Phe Lys Tyr Phe Asp Thr
1315 1320 1325
Thr Ile Asp Arg Lys Arg Tyr Thr Ser Thr Lys Glu Val Leu Asp Ala
1330 1335 1340
Thr Leu Ile His Gln Ser Ile Thr Gly Leu Tyr Glu Thr Arg Ile Asp
1345 1350 1355 1360
Leu Ser Gln Leu Gly Gly Asp
1365
<210> 1333
<211> 1387
<212> Белок
<213> Streptococcus thermophilus
<400> 1333
Thr Lys Pro Tyr Ser Ile Gly Leu Asp Ile Gly Thr Asn Ser Val Gly
1 5 10 15
Trp Ala Val Thr Thr Asp Asn Tyr Lys Val Pro Ser Lys Lys Met Lys
20 25 30
Val Leu Gly Asn Thr Ser Lys Lys Tyr Ile Lys Lys Asn Leu Leu Gly
35 40 45
Val Leu Leu Phe Asp Ser Gly Ile Thr Ala Glu Gly Arg Arg Leu Lys
50 55 60
Arg Thr Ala Arg Arg Arg Tyr Thr Arg Arg Arg Asn Arg Ile Leu Tyr
65 70 75 80
Leu Gln Glu Ile Phe Ser Thr Glu Met Ala Thr Leu Asp Asp Ala Phe
85 90 95
Phe Gln Arg Leu Asp Asp Ser Phe Leu Val Pro Asp Asp Lys Arg Asp
100 105 110
Ser Lys Tyr Pro Ile Phe Gly Asn Leu Val Glu Glu Lys Ala Tyr His
115 120 125
Asp Glu Phe Pro Thr Ile Tyr His Leu Arg Lys Tyr Leu Ala Asp Ser
130 135 140
Thr Lys Lys Ala Asp Leu Arg Leu Val Tyr Leu Ala Leu Ala His Met
145 150 155 160
Ile Lys Tyr Arg Gly His Phe Leu Ile Glu Gly Glu Phe Asn Ser Lys
165 170 175
Asn Asn Asp Ile Gln Lys Asn Phe Gln Asp Phe Leu Asp Thr Tyr Asn
180 185 190
Ala Ile Phe Glu Ser Asp Leu Ser Leu Glu Asn Ser Lys Gln Leu Glu
195 200 205
Glu Ile Val Lys Asp Lys Ile Ser Lys Leu Glu Lys Lys Asp Arg Ile
210 215 220
Leu Lys Leu Phe Pro Gly Glu Lys Asn Ser Gly Ile Phe Ser Glu Phe
225 230 235 240
Leu Lys Leu Ile Val Gly Asn Gln Ala Asp Phe Arg Lys Cys Phe Asn
245 250 255
Leu Asp Glu Lys Ala Ser Leu His Phe Ser Lys Glu Ser Tyr Asp Glu
260 265 270
Asp Leu Glu Thr Leu Leu Gly Tyr Ile Gly Asp Asp Tyr Ser Asp Val
275 280 285
Phe Leu Lys Ala Lys Lys Leu Tyr Asp Ala Ile Leu Leu Ser Gly Phe
290 295 300
Leu Thr Val Thr Asp Asn Glu Thr Glu Ala Pro Leu Ser Ser Ala Met
305 310 315 320
Ile Lys Arg Tyr Asn Glu His Lys Glu Asp Leu Ala Leu Leu Lys Glu
325 330 335
Tyr Ile Arg Asn Ile Ser Leu Lys Thr Tyr Asn Glu Val Phe Lys Asp
340 345 350
Asp Thr Lys Asn Gly Tyr Ala Gly Tyr Ile Asp Gly Lys Thr Asn Gln
355 360 365
Glu Asp Phe Tyr Val Tyr Leu Lys Lys Leu Leu Ala Glu Phe Glu Gly
370 375 380
Ala Asp Tyr Phe Leu Glu Lys Ile Asp Arg Glu Asp Phe Leu Arg Lys
385 390 395 400
Gln Arg Thr Phe Asp Asn Gly Ser Ile Pro Tyr Gln Ile His Leu Gln
405 410 415
Glu Met Arg Ala Ile Leu Asp Lys Gln Ala Lys Phe Tyr Pro Phe Leu
420 425 430
Ala Lys Asn Lys Glu Arg Ile Glu Lys Ile Leu Thr Phe Arg Ile Pro
435 440 445
Tyr Tyr Val Gly Pro Leu Ala Arg Gly Asn Ser Asp Phe Ala Trp Ser
450 455 460
Ile Arg Lys Arg Asn Glu Lys Ile Thr Pro Trp Asn Phe Glu Asp Val
465 470 475 480
Ile Asp Lys Glu Ser Ser Ala Glu Ala Phe Ile Asn Arg Met Thr Ser
485 490 495
Phe Asp Leu Tyr Leu Pro Glu Glu Lys Val Leu Pro Lys His Ser Leu
500 505 510
Leu Tyr Glu Thr Phe Asn Val Tyr Asn Glu Leu Thr Lys Val Arg Phe
515 520 525
Ile Ala Glu Ser Met Arg Asp Tyr Gln Phe Leu Asp Ser Lys Gln Lys
530 535 540
Lys Asp Ile Val Arg Leu Tyr Phe Lys Asp Lys Arg Lys Val Thr Asp
545 550 555 560
Lys Asp Ile Ile Glu Tyr Leu His Ala Ile Tyr Gly Tyr Asp Gly Ile
565 570 575
Glu Leu Lys Gly Ile Glu Lys Gln Phe Asn Ser Ser Leu Ser Thr Tyr
580 585 590
His Asp Leu Leu Asn Ile Ile Asn Asp Lys Glu Phe Leu Asp Asp Ser
595 600 605
Ser Asn Glu Ala Ile Ile Glu Glu Ile Ile His Thr Leu Thr Ile Phe
610 615 620
Glu Asp Arg Glu Met Ile Lys Gln Arg Leu Ser Lys Phe Glu Asn Ile
625 630 635 640
Phe Asp Lys Ser Val Leu Lys Lys Leu Ser Arg Arg His Tyr Thr Gly
645 650 655
Trp Gly Lys Leu Ser Ala Lys Leu Ile Asn Gly Ile Arg Asp Glu Lys
660 665 670
Ser Gly Asn Thr Ile Leu Asp Tyr Leu Ile Asp Asp Gly Ile Ser Asn
675 680 685
Arg Asn Phe Met Gln Leu Ile His Asp Asp Ala Leu Ser Phe Lys Lys
690 695 700
Lys Ile Gln Lys Ala Gln Ile Ile Gly Asp Glu Asp Lys Gly Asn Ile
705 710 715 720
Lys Glu Val Val Lys Ser Leu Pro Gly Ser Pro Ala Ile Lys Lys Gly
725 730 735
Ile Leu Gln Ser Ile Lys Ile Val Asp Glu Leu Val Lys Val Met Gly
740 745 750
Gly Arg Lys Pro Glu Ser Ile Val Val Glu Met Ala Arg Glu Asn Gln
755 760 765
Tyr Thr Asn Gln Gly Lys Ser Asn Ser Gln Gln Arg Leu Lys Arg Leu
770 775 780
Glu Lys Ser Leu Lys Glu Leu Gly Ser Lys Ile Leu Lys Glu Asn Ile
785 790 795 800
Pro Ala Lys Leu Ser Lys Ile Asp Asn Asn Ala Leu Gln Asn Asp Arg
805 810 815
Leu Tyr Leu Tyr Tyr Leu Gln Asn Gly Lys Asp Met Tyr Thr Gly Asp
820 825 830
Asp Leu Asp Ile Asp Arg Leu Ser Asn Tyr Asp Ile Asp His Ile Ile
835 840 845
Pro Gln Ala Phe Leu Lys Asp Asn Ser Ile Asp Asn Lys Val Leu Val
850 855 860
Ser Ser Ala Ser Asn Arg Gly Lys Ser Asp Asp Val Pro Ser Leu Glu
865 870 875 880
Val Val Lys Lys Arg Lys Thr Phe Trp Tyr Gln Leu Leu Lys Ser Lys
885 890 895
Leu Ile Ser Gln Arg Lys Phe Asp Asn Leu Thr Lys Ala Glu Arg Gly
900 905 910
Gly Leu Ser Pro Glu Asp Lys Ala Gly Phe Ile Gln Arg Gln Leu Val
915 920 925
Glu Thr Arg Gln Ile Thr Lys His Val Ala Arg Leu Leu Asp Glu Lys
930 935 940
Phe Asn Asn Lys Lys Asp Glu Asn Asn Arg Ala Val Arg Thr Val Lys
945 950 955 960
Ile Ile Thr Leu Lys Ser Thr Leu Val Ser Gln Phe Arg Lys Asp Phe
965 970 975
Glu Leu Tyr Lys Val Arg Glu Ile Asn Asp Phe His His Ala His Asp
980 985 990
Ala Tyr Leu Asn Ala Val Val Ala Ser Ala Leu Leu Lys Lys Tyr Pro
995 1000 1005
Lys Leu Glu Pro Glu Phe Val Tyr Gly Asp Tyr Pro Lys Tyr Asn Ser
1010 1015 1020
Phe Arg Glu Arg Lys Ser Ala Thr Glu Lys Val Tyr Phe Tyr Ser Asn
1025 1030 1035 1040
Ile Met Asn Ile Phe Lys Lys Ser Ile Ser Leu Ala Asp Gly Arg Val
1045 1050 1055
Ile Glu Arg Pro Leu Ile Glu Val Asn Glu Glu Thr Gly Glu Ser Val
1060 1065 1070
Trp Asn Lys Glu Ser Asp Leu Ala Thr Val Arg Arg Val Leu Ser Tyr
1075 1080 1085
Pro Gln Val Asn Val Val Lys Lys Val Glu Glu Gln Asn His Gly Leu
1090 1095 1100
Asp Arg Gly Lys Pro Lys Gly Leu Phe Asn Ala Asn Leu Ser Ser Lys
1105 1110 1115 1120
Pro Lys Pro Asn Ser Asn Glu Asn Leu Val Gly Ala Lys Glu Tyr Leu
1125 1130 1135
Asp Pro Lys Lys Tyr Gly Gly Tyr Ala Gly Ile Ser Asn Ser Phe Thr
1140 1145 1150
Val Leu Val Lys Gly Thr Ile Glu Lys Gly Ala Lys Lys Lys Ile Thr
1155 1160 1165
Asn Val Leu Glu Phe Gln Gly Ile Ser Ile Leu Asp Arg Ile Asn Tyr
1170 1175 1180
Arg Lys Asp Lys Leu Asn Phe Leu Leu Glu Lys Gly Tyr Lys Asp Ile
1185 1190 1195 1200
Glu Leu Ile Ile Glu Leu Pro Lys Tyr Ser Leu Phe Glu Leu Ser Asp
1205 1210 1215
Gly Ser Arg Arg Met Leu Ala Ser Ile Leu Ser Thr Asn Asn Lys Arg
1220 1225 1230
Gly Glu Ile His Lys Gly Asn Gln Ile Phe Leu Ser Gln Lys Phe Val
1235 1240 1245
Lys Leu Leu Tyr His Ala Lys Arg Ile Ser Asn Thr Ile Asn Glu Asn
1250 1255 1260
His Arg Lys Tyr Val Glu Asn His Lys Lys Glu Phe Glu Glu Leu Phe
1265 1270 1275 1280
Tyr Tyr Ile Leu Glu Phe Asn Glu Asn Tyr Val Gly Ala Lys Lys Asn
1285 1290 1295
Gly Lys Leu Leu Asn Ser Ala Phe Gln Ser Trp Gln Asn His Ser Ile
1300 1305 1310
Asp Glu Leu Cys Ser Ser Phe Ile Gly Pro Thr Gly Ser Glu Arg Lys
1315 1320 1325
Gly Leu Phe Glu Leu Thr Ser Arg Gly Ser Ala Ala Asp Phe Glu Phe
1330 1335 1340
Leu Gly Val Lys Ile Pro Arg Tyr Arg Asp Tyr Thr Pro Ser Ser Leu
1345 1350 1355 1360
Leu Lys Asp Ala Thr Leu Ile His Gln Ser Val Thr Gly Leu Tyr Glu
1365 1370 1375
Thr Arg Ile Asp Leu Ala Lys Leu Gly Glu Gly
1380 1385
<210> 1334
<211> 1333
<212> Белок
<213> Listeria innocua
<400> 1334
Lys Lys Pro Tyr Thr Ile Gly Leu Asp Ile Gly Thr Asn Ser Val Gly
1 5 10 15
Trp Ala Val Leu Thr Asp Gln Tyr Asp Leu Val Lys Arg Lys Met Lys
20 25 30
Ile Ala Gly Asp Ser Glu Lys Lys Gln Ile Lys Lys Asn Phe Trp Gly
35 40 45
Val Arg Leu Phe Asp Glu Gly Gln Thr Ala Ala Asp Arg Arg Met Ala
50 55 60
Arg Thr Ala Arg Arg Arg Ile Glu Arg Arg Arg Asn Arg Ile Ser Tyr
65 70 75 80
Leu Gln Gly Ile Phe Ala Glu Glu Met Ser Lys Thr Asp Ala Asn Phe
85 90 95
Phe Cys Arg Leu Ser Asp Ser Phe Tyr Val Asp Asn Glu Lys Arg Asn
100 105 110
Ser Arg His Pro Phe Phe Ala Thr Ile Glu Glu Glu Val Glu Tyr His
115 120 125
Lys Asn Tyr Pro Thr Ile Tyr His Leu Arg Glu Glu Leu Val Asn Ser
130 135 140
Ser Glu Lys Ala Asp Leu Arg Leu Val Tyr Leu Ala Leu Ala His Ile
145 150 155 160
Ile Lys Tyr Arg Gly Asn Phe Leu Ile Glu Gly Ala Leu Asp Thr Gln
165 170 175
Asn Thr Ser Val Asp Gly Ile Tyr Lys Gln Phe Ile Gln Thr Tyr Asn
180 185 190
Gln Val Phe Ala Ser Gly Ile Glu Asp Gly Ser Leu Lys Lys Leu Glu
195 200 205
Asp Asn Lys Asp Val Ala Lys Ile Leu Val Glu Lys Val Thr Arg Lys
210 215 220
Glu Lys Leu Glu Arg Ile Leu Lys Leu Tyr Pro Gly Glu Lys Ser Ala
225 230 235 240
Gly Met Phe Ala Gln Phe Ile Ser Leu Ile Val Gly Ser Lys Gly Asn
245 250 255
Phe Gln Lys Pro Phe Asp Leu Ile Glu Lys Ser Asp Ile Glu Cys Ala
260 265 270
Lys Asp Ser Tyr Glu Glu Asp Leu Glu Ser Leu Leu Ala Leu Ile Gly
275 280 285
Asp Glu Tyr Ala Glu Leu Phe Val Ala Ala Lys Asn Ala Tyr Ser Ala
290 295 300
Val Val Leu Ser Ser Ile Ile Thr Val Ala Glu Thr Glu Thr Asn Ala
305 310 315 320
Lys Leu Ser Ala Ser Met Ile Glu Arg Phe Asp Thr His Glu Glu Asp
325 330 335
Leu Gly Glu Leu Lys Ala Phe Ile Lys Leu His Leu Pro Lys His Tyr
340 345 350
Glu Glu Ile Phe Ser Asn Thr Glu Lys His Gly Tyr Ala Gly Tyr Ile
355 360 365
Asp Gly Lys Thr Lys Gln Ala Asp Phe Tyr Lys Tyr Met Lys Met Thr
370 375 380
Leu Glu Asn Ile Glu Gly Ala Asp Tyr Phe Ile Ala Lys Ile Glu Lys
385 390 395 400
Glu Asn Phe Leu Arg Lys Gln Arg Thr Phe Asp Asn Gly Ala Ile Pro
405 410 415
His Gln Leu His Leu Glu Glu Leu Glu Ala Ile Leu His Gln Gln Ala
420 425 430
Lys Tyr Tyr Pro Phe Leu Lys Glu Asn Tyr Asp Lys Ile Lys Ser Leu
435 440 445
Val Thr Phe Arg Ile Pro Tyr Phe Val Gly Pro Leu Ala Asn Gly Gln
450 455 460
Ser Glu Phe Ala Trp Leu Thr Arg Lys Ala Asp Gly Glu Ile Arg Pro
465 470 475 480
Trp Asn Ile Glu Glu Lys Val Asp Phe Gly Lys Ser Ala Val Asp Phe
485 490 495
Ile Glu Lys Met Thr Asn Lys Asp Thr Tyr Leu Pro Lys Glu Asn Val
500 505 510
Leu Pro Lys His Ser Leu Cys Tyr Gln Lys Tyr Leu Val Tyr Asn Glu
515 520 525
Leu Thr Lys Val Arg Tyr Ile Asn Asp Gln Gly Lys Thr Ser Tyr Phe
530 535 540
Ser Gly Gln Glu Lys Glu Gln Ile Phe Asn Asp Leu Phe Lys Gln Lys
545 550 555 560
Arg Lys Val Lys Lys Lys Asp Leu Glu Leu Phe Leu Arg Asn Met Ser
565 570 575
His Val Glu Ser Pro Thr Ile Glu Gly Leu Glu Asp Ser Phe Asn Ser
580 585 590
Ser Tyr Ser Thr Tyr His Asp Leu Leu Lys Val Gly Ile Lys Gln Glu
595 600 605
Ile Leu Asp Asn Pro Val Asn Thr Glu Met Leu Glu Asn Ile Val Lys
610 615 620
Ile Leu Thr Val Phe Glu Asp Lys Arg Met Ile Lys Glu Gln Leu Gln
625 630 635 640
Gln Phe Ser Asp Val Leu Asp Gly Val Val Leu Lys Lys Leu Glu Arg
645 650 655
Arg His Tyr Thr Gly Trp Gly Arg Leu Ser Ala Lys Leu Leu Met Gly
660 665 670
Ile Arg Asp Lys Gln Ser His Leu Thr Ile Leu Asp Tyr Leu Met Asn
675 680 685
Asp Asp Gly Leu Asn Arg Asn Leu Met Gln Leu Ile Asn Asp Ser Asn
690 695 700
Leu Ser Phe Lys Ser Ile Ile Glu Lys Glu Gln Val Thr Thr Ala Asp
705 710 715 720
Lys Asp Ile Gln Ser Ile Val Ala Asp Leu Ala Gly Ser Pro Ala Ile
725 730 735
Lys Lys Gly Ile Leu Gln Ser Leu Lys Ile Val Asp Glu Leu Val Ser
740 745 750
Val Met Gly Tyr Pro Pro Gln Thr Ile Val Val Glu Met Ala Arg Glu
755 760 765
Asn Gln Thr Thr Gly Lys Gly Lys Asn Asn Ser Arg Pro Arg Tyr Lys
770 775 780
Ser Leu Glu Lys Ala Ile Lys Glu Phe Gly Ser Gln Ile Leu Lys Glu
785 790 795 800
His Pro Thr Asp Asn Gln Glu Leu Arg Asn Asn Arg Leu Tyr Leu Tyr
805 810 815
Tyr Leu Gln Asn Gly Lys Asp Met Tyr Thr Gly Gln Asp Leu Asp Ile
820 825 830
His Asn Leu Ser Asn Tyr Asp Ile Asp His Ile Val Pro Gln Ser Phe
835 840 845
Ile Thr Asp Asn Ser Ile Asp Asn Leu Val Leu Thr Ser Ser Ala Gly
850 855 860
Asn Arg Glu Lys Gly Asp Asp Val Pro Pro Leu Glu Ile Val Arg Lys
865 870 875 880
Arg Lys Val Phe Trp Glu Lys Leu Tyr Gln Gly Asn Leu Met Ser Lys
885 890 895
Arg Lys Phe Asp Tyr Leu Thr Lys Ala Glu Arg Gly Gly Leu Thr Glu
900 905 910
Ala Asp Lys Ala Arg Phe Ile His Arg Gln Leu Val Glu Thr Arg Gln
915 920 925
Ile Thr Lys Asn Val Ala Asn Ile Leu His Gln Arg Phe Asn Tyr Glu
930 935 940
Lys Asp Asp His Gly Asn Thr Met Lys Gln Val Arg Ile Val Thr Leu
945 950 955 960
Lys Ser Ala Leu Val Ser Gln Phe Arg Lys Gln Phe Gln Leu Tyr Lys
965 970 975
Val Arg Asp Val Asn Asp Tyr His His Ala His Asp Ala Tyr Leu Asn
980 985 990
Gly Val Val Ala Asn Thr Leu Leu Lys Val Tyr Pro Gln Leu Glu Pro
995 1000 1005
Glu Phe Val Tyr Gly Asp Tyr His Gln Phe Asp Trp Phe Lys Ala Asn
1010 1015 1020
Lys Ala Thr Ala Lys Lys Gln Phe Tyr Thr Asn Ile Met Leu Phe Phe
1025 1030 1035 1040
Ala Gln Lys Asp Arg Ile Ile Asp Glu Asn Gly Glu Ile Leu Trp Asp
1045 1050 1055
Lys Lys Tyr Leu Asp Thr Val Lys Lys Val Met Ser Tyr Arg Gln Met
1060 1065 1070
Asn Ile Val Lys Lys Thr Glu Ile Gln Lys Gly Glu Phe Ser Lys Ala
1075 1080 1085
Thr Ile Lys Pro Lys Gly Asn Ser Ser Lys Leu Ile Pro Arg Lys Thr
1090 1095 1100
Asn Trp Asp Pro Met Lys Tyr Gly Gly Leu Asp Ser Pro Asn Met Ala
1105 1110 1115 1120
Tyr Ala Val Val Ile Glu Tyr Ala Lys Gly Lys Asn Lys Leu Val Phe
1125 1130 1135
Glu Lys Lys Ile Ile Arg Val Thr Ile Met Glu Arg Lys Ala Phe Glu
1140 1145 1150
Lys Asp Glu Lys Ala Phe Leu Glu Glu Gln Gly Tyr Arg Gln Pro Lys
1155 1160 1165
Val Leu Ala Lys Leu Pro Lys Tyr Thr Leu Tyr Glu Cys Glu Glu Gly
1170 1175 1180
Arg Arg Arg Met Leu Ala Ser Ala Asn Glu Ala Gln Lys Gly Asn Gln
1185 1190 1195 1200
Gln Val Leu Pro Asn His Leu Val Thr Leu Leu His His Ala Ala Asn
1205 1210 1215
Cys Glu Val Ser Asp Gly Lys Ser Leu Asp Tyr Ile Glu Ser Asn Arg
1220 1225 1230
Glu Met Phe Ala Glu Leu Leu Ala His Val Ser Glu Phe Ala Lys Arg
1235 1240 1245
Tyr Thr Leu Ala Glu Ala Asn Leu Asn Lys Ile Asn Gln Leu Phe Glu
1250 1255 1260
Gln Asn Lys Glu Gly Asp Ile Lys Ala Ile Ala Gln Ser Phe Val Asp
1265 1270 1275 1280
Leu Met Ala Phe Asn Ala Met Gly Ala Pro Ala Ser Phe Lys Phe Phe
1285 1290 1295
Glu Thr Thr Ile Glu Arg Lys Arg Tyr Asn Asn Leu Lys Glu Leu Leu
1300 1305 1310
Asn Ser Thr Ile Ile Tyr Gln Ser Ile Thr Gly Leu Tyr Glu Ser Arg
1315 1320 1325
Lys Arg Leu Asp Asp
1330
<210> 1335
<211> 1082
<212> Белок
<213> Neisseria meningitidis
<400> 1335
Met Ala Ala Phe Lys Pro Asn Ser Ile Asn Tyr Ile Leu Gly Leu Asp
1 5 10 15
Ile Gly Ile Ala Ser Val Gly Trp Ala Met Val Glu Ile Asp Glu Glu
20 25 30
Glu Asn Pro Ile Arg Leu Ile Asp Leu Gly Val Arg Val Phe Glu Arg
35 40 45
Ala Glu Val Pro Lys Thr Gly Asp Ser Leu Ala Met Ala Arg Arg Leu
50 55 60
Ala Arg Ser Val Arg Arg Leu Thr Arg Arg Arg Ala His Arg Leu Leu
65 70 75 80
Arg Thr Arg Arg Leu Leu Lys Arg Glu Gly Val Leu Gln Ala Ala Asn
85 90 95
Phe Asp Glu Asn Gly Leu Ile Lys Ser Leu Pro Asn Thr Pro Trp Gln
100 105 110
Leu Arg Ala Ala Ala Leu Asp Arg Lys Leu Thr Pro Leu Glu Trp Ser
115 120 125
Ala Val Leu Leu His Leu Ile Lys His Arg Gly Tyr Leu Ser Gln Arg
130 135 140
Lys Asn Glu Gly Glu Thr Ala Asp Lys Glu Leu Gly Ala Leu Leu Lys
145 150 155 160
Gly Val Ala Gly Asn Ala His Ala Leu Gln Thr Gly Asp Phe Arg Thr
165 170 175
Pro Ala Glu Leu Ala Leu Asn Lys Phe Glu Lys Glu Ser Gly His Ile
180 185 190
Arg Asn Gln Arg Ser Asp Tyr Ser His Thr Phe Ser Arg Lys Asp Leu
195 200 205
Gln Ala Glu Leu Ile Leu Leu Phe Glu Lys Gln Lys Glu Phe Gly Asn
210 215 220
Pro His Val Ser Gly Gly Leu Lys Glu Gly Ile Glu Thr Leu Leu Met
225 230 235 240
Thr Gln Arg Pro Ala Leu Ser Gly Asp Ala Val Gln Lys Met Leu Gly
245 250 255
His Cys Thr Phe Glu Pro Ala Glu Pro Lys Ala Ala Lys Asn Thr Tyr
260 265 270
Thr Ala Glu Arg Phe Ile Trp Leu Thr Lys Leu Asn Asn Leu Arg Ile
275 280 285
Leu Glu Gln Gly Ser Glu Arg Pro Leu Thr Asp Thr Glu Arg Ala Thr
290 295 300
Leu Met Asp Glu Pro Tyr Arg Lys Ser Lys Leu Thr Tyr Ala Gln Ala
305 310 315 320
Arg Lys Leu Leu Gly Leu Glu Asp Thr Ala Phe Phe Lys Gly Leu Arg
325 330 335
Tyr Gly Lys Asp Asn Ala Glu Ala Ser Thr Leu Met Glu Met Lys Ala
340 345 350
Tyr His Ala Ile Ser Arg Ala Leu Glu Lys Glu Gly Leu Lys Asp Lys
355 360 365
Lys Ser Pro Leu Asn Leu Ser Pro Glu Leu Gln Asp Glu Ile Gly Thr
370 375 380
Ala Phe Ser Leu Phe Lys Thr Asp Glu Asp Ile Thr Gly Arg Leu Lys
385 390 395 400
Asp Arg Ile Gln Pro Glu Ile Leu Glu Ala Leu Leu Lys His Ile Ser
405 410 415
Phe Asp Lys Phe Val Gln Ile Ser Leu Lys Ala Leu Arg Arg Ile Val
420 425 430
Pro Leu Met Glu Gln Gly Lys Arg Tyr Asp Glu Ala Cys Ala Glu Ile
435 440 445
Tyr Gly Asp His Tyr Gly Lys Lys Asn Thr Glu Glu Lys Ile Tyr Leu
450 455 460
Pro Pro Ile Pro Ala Asp Glu Ile Arg Asn Pro Val Val Leu Arg Ala
465 470 475 480
Leu Ser Gln Ala Arg Lys Val Ile Asn Gly Val Val Arg Arg Tyr Gly
485 490 495
Ser Pro Ala Arg Ile His Ile Glu Thr Ala Arg Glu Val Gly Lys Ser
500 505 510
Phe Lys Asp Arg Lys Glu Ile Glu Lys Arg Gln Glu Glu Asn Arg Lys
515 520 525
Asp Arg Glu Lys Ala Ala Ala Lys Phe Arg Glu Tyr Phe Pro Asn Phe
530 535 540
Val Gly Glu Pro Lys Ser Lys Asp Ile Leu Lys Leu Arg Leu Tyr Glu
545 550 555 560
Gln Gln His Gly Lys Cys Leu Tyr Ser Gly Lys Glu Ile Asn Leu Gly
565 570 575
Arg Leu Asn Glu Lys Gly Tyr Val Glu Ile Asp His Ala Leu Pro Phe
580 585 590
Ser Arg Thr Trp Asp Asp Ser Phe Asn Asn Lys Val Leu Val Leu Gly
595 600 605
Ser Glu Asn Gln Asn Lys Gly Asn Gln Thr Pro Tyr Glu Tyr Phe Asn
610 615 620
Gly Lys Asp Asn Ser Arg Glu Trp Gln Glu Phe Lys Ala Arg Val Glu
625 630 635 640
Thr Ser Arg Phe Pro Arg Ser Lys Lys Gln Arg Ile Leu Leu Gln Lys
645 650 655
Phe Asp Glu Asp Gly Phe Lys Glu Arg Asn Leu Asn Asp Thr Arg Tyr
660 665 670
Val Asn Arg Phe Leu Cys Gln Phe Val Ala Asp Arg Met Arg Leu Thr
675 680 685
Gly Lys Gly Lys Lys Arg Val Phe Ala Ser Asn Gly Gln Ile Thr Asn
690 695 700
Leu Leu Arg Gly Phe Trp Gly Leu Arg Lys Val Arg Ala Glu Asn Asp
705 710 715 720
Arg His His Ala Leu Asp Ala Val Val Val Ala Cys Ser Thr Val Ala
725 730 735
Met Gln Gln Lys Ile Thr Arg Phe Val Arg Tyr Lys Glu Met Asn Ala
740 745 750
Phe Asp Gly Lys Thr Ile Asp Lys Glu Thr Gly Glu Val Leu His Gln
755 760 765
Lys Thr His Phe Pro Gln Pro Trp Glu Phe Phe Ala Gln Glu Val Met
770 775 780
Ile Arg Val Phe Gly Lys Pro Asp Gly Lys Pro Glu Phe Glu Glu Ala
785 790 795 800
Asp Thr Leu Glu Lys Leu Arg Thr Leu Leu Ala Glu Lys Leu Ser Ser
805 810 815
Arg Pro Glu Ala Val His Glu Tyr Val Thr Pro Leu Phe Val Ser Arg
820 825 830
Ala Pro Asn Arg Lys Met Ser Gly Gln Gly His Met Glu Thr Val Lys
835 840 845
Ser Ala Lys Arg Leu Asp Glu Gly Val Ser Val Leu Arg Val Pro Leu
850 855 860
Thr Gln Leu Lys Leu Lys Asp Leu Glu Lys Met Val Asn Arg Glu Arg
865 870 875 880
Glu Pro Lys Leu Tyr Glu Ala Leu Lys Ala Arg Leu Glu Ala His Lys
885 890 895
Asp Asp Pro Ala Lys Ala Phe Ala Glu Pro Phe Tyr Lys Tyr Asp Lys
900 905 910
Ala Gly Asn Arg Thr Gln Gln Val Lys Ala Val Arg Val Glu Gln Val
915 920 925
Gln Lys Thr Gly Val Trp Val Arg Asn His Asn Gly Ile Ala Asp Asn
930 935 940
Ala Thr Met Val Arg Val Asp Val Phe Glu Lys Gly Asp Lys Tyr Tyr
945 950 955 960
Leu Val Pro Ile Tyr Ser Trp Gln Val Ala Lys Gly Ile Leu Pro Asp
965 970 975
Arg Ala Val Val Gln Gly Lys Asp Glu Glu Asp Trp Gln Leu Ile Asp
980 985 990
Asp Ser Phe Asn Phe Lys Phe Ser Leu His Pro Asn Asp Leu Val Glu
995 1000 1005
Val Ile Thr Lys Lys Ala Arg Met Phe Gly Tyr Phe Ala Ser Cys His
1010 1015 1020
Arg Gly Thr Gly Asn Ile Asn Ile Arg Ile His Asp Leu Asp His Lys
1025 1030 1035 1040
Ile Gly Lys Asn Gly Ile Leu Glu Gly Ile Gly Val Lys Thr Ala Leu
1045 1050 1055
Ser Phe Gln Lys Tyr Gln Ile Asp Glu Leu Gly Lys Glu Ile Arg Pro
1060 1065 1070
Cys Arg Leu Lys Lys Arg Pro Pro Val Arg
1075 1080
<210> 1336
<211> 1368
<212> Белок
<213> Streptococcus pyogenes
<400> 1336
Met Asp Lys Lys Tyr Ser Ile Gly Leu Asp Ile Gly Thr Asn Ser Val
1 5 10 15
Gly Trp Ala Val Ile Thr Asp Glu Tyr Lys Val Pro Ser Lys Lys Phe
20 25 30
Lys Val Leu Gly Asn Thr Asp Arg His Ser Ile Lys Lys Asn Leu Ile
35 40 45
Gly Ala Leu Leu Phe Asp Ser Gly Glu Thr Ala Glu Ala Thr Arg Leu
50 55 60
Lys Arg Thr Ala Arg Arg Arg Tyr Thr Arg Arg Lys Asn Arg Ile Cys
65 70 75 80
Tyr Leu Gln Glu Ile Phe Ser Asn Glu Met Ala Lys Val Asp Asp Ser
85 90 95
Phe Phe His Arg Leu Glu Glu Ser Phe Leu Val Glu Glu Asp Lys Lys
100 105 110
His Glu Arg His Pro Ile Phe Gly Asn Ile Val Asp Glu Val Ala Tyr
115 120 125
His Glu Lys Tyr Pro Thr Ile Tyr His Leu Arg Lys Lys Leu Val Asp
130 135 140
Ser Thr Asp Lys Ala Asp Leu Arg Leu Ile Tyr Leu Ala Leu Ala His
145 150 155 160
Met Ile Lys Phe Arg Gly His Phe Leu Ile Glu Gly Asp Leu Asn Pro
165 170 175
Asp Asn Ser Asp Val Asp Lys Leu Phe Ile Gln Leu Val Gln Thr Tyr
180 185 190
Asn Gln Leu Phe Glu Glu Asn Pro Ile Asn Ala Ser Gly Val Asp Ala
195 200 205
Lys Ala Ile Leu Ser Ala Arg Leu Ser Lys Ser Arg Arg Leu Glu Asn
210 215 220
Leu Ile Ala Gln Leu Pro Gly Glu Lys Lys Asn Gly Leu Phe Gly Asn
225 230 235 240
Leu Ile Ala Leu Ser Leu Gly Leu Thr Pro Asn Phe Lys Ser Asn Phe
245 250 255
Asp Leu Ala Glu Asp Ala Lys Leu Gln Leu Ser Lys Asp Thr Tyr Asp
260 265 270
Asp Asp Leu Asp Asn Leu Leu Ala Gln Ile Gly Asp Gln Tyr Ala Asp
275 280 285
Leu Phe Leu Ala Ala Lys Asn Leu Ser Asp Ala Ile Leu Leu Ser Asp
290 295 300
Ile Leu Arg Val Asn Thr Glu Ile Thr Lys Ala Pro Leu Ser Ala Ser
305 310 315 320
Met Ile Lys Arg Tyr Asp Glu His His Gln Asp Leu Thr Leu Leu Lys
325 330 335
Ala Leu Val Arg Gln Gln Leu Pro Glu Lys Tyr Lys Glu Ile Phe Phe
340 345 350
Asp Gln Ser Lys Asn Gly Tyr Ala Gly Tyr Ile Asp Gly Gly Ala Ser
355 360 365
Gln Glu Glu Phe Tyr Lys Phe Ile Lys Pro Ile Leu Glu Lys Met Asp
370 375 380
Gly Thr Glu Glu Leu Leu Val Lys Leu Asn Arg Glu Asp Leu Leu Arg
385 390 395 400
Lys Gln Arg Thr Phe Asp Asn Gly Ser Ile Pro His Gln Ile His Leu
405 410 415
Gly Glu Leu His Ala Ile Leu Arg Arg Gln Glu Asp Phe Tyr Pro Phe
420 425 430
Leu Lys Asp Asn Arg Glu Lys Ile Glu Lys Ile Leu Thr Phe Arg Ile
435 440 445
Pro Tyr Tyr Val Gly Pro Leu Ala Arg Gly Asn Ser Arg Phe Ala Trp
450 455 460
Met Thr Arg Lys Ser Glu Glu Thr Ile Thr Pro Trp Asn Phe Glu Glu
465 470 475 480
Val Val Asp Lys Gly Ala Ser Ala Gln Ser Phe Ile Glu Arg Met Thr
485 490 495
Asn Phe Asp Lys Asn Leu Pro Asn Glu Lys Val Leu Pro Lys His Ser
500 505 510
Leu Leu Tyr Glu Tyr Phe Thr Val Tyr Asn Glu Leu Thr Lys Val Lys
515 520 525
Tyr Val Thr Glu Gly Met Arg Lys Pro Ala Phe Leu Ser Gly Glu Gln
530 535 540
Lys Lys Ala Ile Val Asp Leu Leu Phe Lys Thr Asn Arg Lys Val Thr
545 550 555 560
Val Lys Gln Leu Lys Glu Asp Tyr Phe Lys Lys Ile Glu Cys Phe Asp
565 570 575
Ser Val Glu Ile Ser Gly Val Glu Asp Arg Phe Asn Ala Ser Leu Gly
580 585 590
Thr Tyr His Asp Leu Leu Lys Ile Ile Lys Asp Lys Asp Phe Leu Asp
595 600 605
Asn Glu Glu Asn Glu Asp Ile Leu Glu Asp Ile Val Leu Thr Leu Thr
610 615 620
Leu Phe Glu Asp Arg Glu Met Ile Glu Glu Arg Leu Lys Thr Tyr Ala
625 630 635 640
His Leu Phe Asp Asp Lys Val Met Lys Gln Leu Lys Arg Arg Arg Tyr
645 650 655
Thr Gly Trp Gly Arg Leu Ser Arg Lys Leu Ile Asn Gly Ile Arg Asp
660 665 670
Lys Gln Ser Gly Lys Thr Ile Leu Asp Phe Leu Lys Ser Asp Gly Phe
675 680 685
Ala Asn Arg Asn Phe Met Gln Leu Ile His Asp Asp Ser Leu Thr Phe
690 695 700
Lys Glu Asp Ile Gln Lys Ala Gln Val Ser Gly Gln Gly Asp Ser Leu
705 710 715 720
His Glu His Ile Ala Asn Leu Ala Gly Ser Pro Ala Ile Lys Lys Gly
725 730 735
Ile Leu Gln Thr Val Lys Val Val Asp Glu Leu Val Lys Val Met Gly
740 745 750
Arg His Lys Pro Glu Asn Ile Val Ile Glu Met Ala Arg Glu Asn Gln
755 760 765
Thr Thr Gln Lys Gly Gln Lys Asn Ser Arg Glu Arg Met Lys Arg Ile
770 775 780
Glu Glu Gly Ile Lys Glu Leu Gly Ser Gln Ile Leu Lys Glu His Pro
785 790 795 800
Val Glu Asn Thr Gln Leu Gln Asn Glu Lys Leu Tyr Leu Tyr Tyr Leu
805 810 815
Gln Asn Gly Arg Asp Met Tyr Val Asp Gln Glu Leu Asp Ile Asn Arg
820 825 830
Leu Ser Asp Tyr Asp Val Asp His Ile Val Pro Gln Ser Phe Leu Lys
835 840 845
Asp Asp Ser Ile Asp Asn Lys Val Leu Thr Arg Ser Asp Lys Asn Arg
850 855 860
Gly Lys Ser Asp Asn Val Pro Ser Glu Glu Val Val Lys Lys Met Lys
865 870 875 880
Asn Tyr Trp Arg Gln Leu Leu Asn Ala Lys Leu Ile Thr Gln Arg Lys
885 890 895
Phe Asp Asn Leu Thr Lys Ala Glu Arg Gly Gly Leu Ser Glu Leu Asp
900 905 910
Lys Ala Gly Phe Ile Lys Arg Gln Leu Val Glu Thr Arg Gln Ile Thr
915 920 925
Lys His Val Ala Gln Ile Leu Asp Ser Arg Met Asn Thr Lys Tyr Asp
930 935 940
Glu Asn Asp Lys Leu Ile Arg Glu Val Lys Val Ile Thr Leu Lys Ser
945 950 955 960
Lys Leu Val Ser Asp Phe Arg Lys Asp Phe Gln Phe Tyr Lys Val Arg
965 970 975
Glu Ile Asn Asn Tyr His His Ala His Asp Ala Tyr Leu Asn Ala Val
980 985 990
Val Gly Thr Ala Leu Ile Lys Lys Tyr Pro Lys Leu Glu Ser Glu Phe
995 1000 1005
Val Tyr Gly Asp Tyr Lys Val Tyr Asp Val Arg Lys Met Ile Ala Lys
1010 1015 1020
Ser Glu Gln Glu Ile Gly Lys Ala Thr Ala Lys Tyr Phe Phe Tyr Ser
1025 1030 1035 1040
Asn Ile Met Asn Phe Phe Lys Thr Glu Ile Thr Leu Ala Asn Gly Glu
1045 1050 1055
Ile Arg Lys Arg Pro Leu Ile Glu Thr Asn Gly Glu Thr Gly Glu Ile
1060 1065 1070
Val Trp Asp Lys Gly Arg Asp Phe Ala Thr Val Arg Lys Val Leu Ser
1075 1080 1085
Met Pro Gln Val Asn Ile Val Lys Lys Thr Glu Val Gln Thr Gly Gly
1090 1095 1100
Phe Ser Lys Glu Ser Ile Leu Pro Lys Arg Asn Ser Asp Lys Leu Ile
1105 1110 1115 1120
Ala Arg Lys Lys Asp Trp Asp Pro Lys Lys Tyr Gly Gly Phe Asp Ser
1125 1130 1135
Pro Thr Val Ala Tyr Ser Val Leu Val Val Ala Lys Val Glu Lys Gly
1140 1145 1150
Lys Ser Lys Lys Leu Lys Ser Val Lys Glu Leu Leu Gly Ile Thr Ile
1155 1160 1165
Met Glu Arg Ser Ser Phe Glu Lys Asn Pro Ile Asp Phe Leu Glu Ala
1170 1175 1180
Lys Gly Tyr Lys Glu Val Lys Lys Asp Leu Ile Ile Lys Leu Pro Lys
1185 1190 1195 1200
Tyr Ser Leu Phe Glu Leu Glu Asn Gly Arg Lys Arg Met Leu Ala Ser
1205 1210 1215
Ala Gly Glu Leu Gln Lys Gly Asn Glu Leu Ala Leu Pro Ser Lys Tyr
1220 1225 1230
Val Asn Phe Leu Tyr Leu Ala Ser His Tyr Glu Lys Leu Lys Gly Ser
1235 1240 1245
Pro Glu Asp Asn Glu Gln Lys Gln Leu Phe Val Glu Gln His Lys His
1250 1255 1260
Tyr Leu Asp Glu Ile Ile Glu Gln Ile Ser Glu Phe Ser Lys Arg Val
1265 1270 1275 1280
Ile Leu Ala Asp Ala Asn Leu Asp Lys Val Leu Ser Ala Tyr Asn Lys
1285 1290 1295
His Arg Asp Lys Pro Ile Arg Glu Gln Ala Glu Asn Ile Ile His Leu
1300 1305 1310
Phe Thr Leu Thr Asn Leu Gly Ala Pro Ala Ala Phe Lys Tyr Phe Asp
1315 1320 1325
Thr Thr Ile Asp Arg Lys Arg Tyr Thr Ser Thr Lys Glu Val Leu Asp
1330 1335 1340
Ala Thr Leu Ile His Gln Ser Ile Thr Gly Leu Tyr Glu Thr Arg Ile
1345 1350 1355 1360
Asp Leu Ser Gln Leu Gly Gly Asp
1365
<210> 1337
<211> 4107
<212> ДНК
<213> Искусственная последовательность
<220>
<223> S. pyogenes Cas9, кодон-оптимизированная последовательность нуклеиновой кислоты
<400> 1337
atggataaaa agtacagcat cgggctggac atcggtacaa actcagtggg gtgggccgtg 60
attacggacg agtacaaggt accctccaaa aaatttaaag tgctgggtaa cacggacaga 120
cactctataa agaaaaatct tattggagcc ttgctgttcg actcaggcga gacagccgaa 180
gccacaaggt tgaagcggac cgccaggagg cggtatacca ggagaaagaa ccgcatatgc 240
tacctgcaag aaatcttcag taacgagatg gcaaaggttg acgatagctt tttccatcgc 300
ctggaagaat cctttcttgt tgaggaagac aagaagcacg aacggcaccc catctttggc 360
aatattgtcg acgaagtggc atatcacgaa aagtacccga ctatctacca cctcaggaag 420
aagctggtgg actctaccga taaggcggac ctcagactta tttatttggc actcgcccac 480
atgattaaat ttagaggaca tttcttgatc gagggcgacc tgaacccgga caacagtgac 540
gtcgataagc tgttcatcca acttgtgcag acctacaatc aactgttcga agaaaaccct 600
ataaatgctt caggagtcga cgctaaagca atcctgtccg cgcgcctctc aaaatctaga 660
agacttgaga atctgattgc tcagttgccc ggggaaaaga aaaatggatt gtttggcaac 720
ctgatcgccc tcagtctcgg actgacccca aatttcaaaa gtaacttcga cctggccgaa 780
gacgctaagc tccagctgtc caaggacaca tacgatgacg acctcgacaa tctgctggcc 840
cagattgggg atcagtacgc cgatctcttt ttggcagcaa agaacctgtc cgacgccatc 900
ctgttgagcg atatcttgag agtgaacacc gaaattacta aagcacccct tagcgcatct 960
atgatcaagc ggtacgacga gcatcatcag gatctgaccc tgctgaaggc tcttgtgagg 1020
caacagctcc ccgaaaaata caaggaaatc ttctttgacc agagcaaaaa cggctacgct 1080
ggctatatag atggtggggc cagtcaggag gaattctata aattcatcaa gcccattctc 1140
gagaaaatgg acggcacaga ggagttgctg gtcaaactta acagggagga cctgctgcgg 1200
aagcagcgga cctttgacaa cgggtctatc ccccaccaga ttcatctggg cgaactgcac 1260
gcaatcctga ggaggcagga ggatttttat ccttttctta aagataaccg cgagaaaata 1320
gaaaagattc ttacattcag gatcccgtac tacgtgggac ctctcgcccg gggcaattca 1380
cggtttgcct ggatgacaag gaagtcagag gagactatta caccttggaa cttcgaagaa 1440
gtggtggaca agggtgcatc tgcccagtct ttcatcgagc ggatgacaaa ttttgacaag 1500
aacctcccta atgagaaggt gctgcccaaa cattctctgc tctacgagta ctttaccgtc 1560
tacaatgaac tgactaaagt caagtacgtc accgagggaa tgaggaagcc ggcattcctt 1620
agtggagaac agaagaaggc gattgtagac ctgttgttca agaccaacag gaaggtgact 1680
gtgaagcaac ttaaagaaga ctactttaag aagatcgaat gttttgacag tgtggaaatt 1740
tcaggggttg aagaccgctt caatgcgtca ttggggactt accatgatct tctcaagatc 1800
ataaaggaca aagacttcct ggacaacgaa gaaaatgagg atattctcga agacatcgtc 1860
ctcaccctga ccctgttcga agacagggaa atgatagaag agcgcttgaa aacctatgcc 1920
cacctcttcg acgataaagt tatgaagcag ctgaagcgca ggagatacac aggatgggga 1980
agattgtcaa ggaagctgat caatggaatt agggataaac agagtggcaa gaccatactg 2040
gatttcctca aatctgatgg cttcgccaat aggaacttca tgcaactgat tcacgatgac 2100
tctcttacct tcaaggagga cattcaaaag gctcaggtga gcgggcaggg agactccctt 2160
catgaacaca tcgcgaattt ggcaggttcc cccgctatta aaaagggcat ccttcaaact 2220
gtcaaggtgg tggatgaatt ggtcaaggta atgggcagac ataagccaga aaatattgtg 2280
atcgagatgg cccgcgaaaa ccagaccaca cagaagggcc agaaaaatag tagagagcgg 2340
atgaagagga tcgaggaggg catcaaagag ctgggatctc agattctcaa agaacacccc 2400
gtagaaaaca cacagctgca gaacgaaaaa ttgtacttgt actatctgca gaacggcaga 2460
gacatgtacg tcgaccaaga acttgatatt aatagactgt ccgactatga cgtagaccat 2520
atcgtgcccc agtccttcct gaaggacgac tccattgata acaaagtctt gacaagaagc 2580
gacaagaaca ggggtaaaag tgataatgtg cctagcgagg aggtggtgaa aaaaatgaag 2640
aactactggc gacagctgct taatgcaaag ctcattacac aacggaagtt cgataatctg 2700
acgaaagcag agagaggtgg cttgtctgag ttggacaagg cagggtttat taagcggcag 2760
ctggtggaaa ctaggcagat cacaaagcac gtggcgcaga ttttggacag ccggatgaac 2820
acaaaatacg acgaaaatga taaactgata cgagaggtca aagttatcac gctgaaaagc 2880
aagctggtgt ccgattttcg gaaagacttc cagttctaca aagttcgcga gattaataac 2940
taccatcatg ctcacgatgc gtacctgaac gctgttgtcg ggaccgcctt gataaagaag 3000
tacccaaagc tggaatccga gttcgtatac ggggattaca aagtgtacga tgtgaggaaa 3060
atgatagcca agtccgagca ggagattgga aaggccacag ctaagtactt cttttattct 3120
aacatcatga atttttttaa gacggaaatt accctggcca acggagagat cagaaagcgg 3180
ccccttatag agacaaatgg tgaaacaggt gaaatcgtct gggataaggg cagggatttc 3240
gctactgtga ggaaggtgct gagtatgcca caggtaaata tcgtgaaaaa aaccgaagta 3300
cagaccggag gattttccaa ggaaagcatt ttgcctaaaa gaaactcaga caagctcatc 3360
gcccgcaaga aagattggga ccctaagaaa tacgggggat ttgactcacc caccgtagcc 3420
tattctgtgc tggtggtagc taaggtggaa aaaggaaagt ctaagaagct gaagtccgtg 3480
aaggaactct tgggaatcac tatcatggaa agatcatcct ttgaaaagaa ccctatcgat 3540
ttcctggagg ctaagggtta caaggaggtc aagaaagacc tcatcattaa actgccaaaa 3600
tactctctct tcgagctgga aaatggcagg aagagaatgt tggccagcgc cggagagctg 3660
caaaagggaa acgagcttgc tctgccctcc aaatatgtta attttctcta tctcgcttcc 3720
cactatgaaa agctgaaagg gtctcccgaa gataacgagc agaagcagct gttcgtcgaa 3780
cagcacaagc actatctgga tgaaataatc gaacaaataa gcgagttcag caaaagggtt 3840
atcctggcgg atgctaattt ggacaaagta ctgtctgctt ataacaagca ccgggataag 3900
cctattaggg aacaagccga gaatataatt cacctcttta cactcacgaa tctcggagcc 3960
cccgccgcct tcaaatactt tgatacgact atcgaccgga aacggtatac cagtaccaaa 4020
gaggtcctcg atgccaccct catccaccag tcaattactg gcctgtacga aacacggatc 4080
gacctctctc aactgggcgg cgactag 4107
<210> 1338
<211> 1368
<212> Белок
<213> Streptococcus pyogenes
<400> 1338
Met Asp Lys Lys Tyr Ser Ile Gly Leu Asp Ile Gly Thr Asn Ser Val
1 5 10 15
Gly Trp Ala Val Ile Thr Asp Glu Tyr Lys Val Pro Ser Lys Lys Phe
20 25 30
Lys Val Leu Gly Asn Thr Asp Arg His Ser Ile Lys Lys Asn Leu Ile
35 40 45
Gly Ala Leu Leu Phe Asp Ser Gly Glu Thr Ala Glu Ala Thr Arg Leu
50 55 60
Lys Arg Thr Ala Arg Arg Arg Tyr Thr Arg Arg Lys Asn Arg Ile Cys
65 70 75 80
Tyr Leu Gln Glu Ile Phe Ser Asn Glu Met Ala Lys Val Asp Asp Ser
85 90 95
Phe Phe His Arg Leu Glu Glu Ser Phe Leu Val Glu Glu Asp Lys Lys
100 105 110
His Glu Arg His Pro Ile Phe Gly Asn Ile Val Asp Glu Val Ala Tyr
115 120 125
His Glu Lys Tyr Pro Thr Ile Tyr His Leu Arg Lys Lys Leu Val Asp
130 135 140
Ser Thr Asp Lys Ala Asp Leu Arg Leu Ile Tyr Leu Ala Leu Ala His
145 150 155 160
Met Ile Lys Phe Arg Gly His Phe Leu Ile Glu Gly Asp Leu Asn Pro
165 170 175
Asp Asn Ser Asp Val Asp Lys Leu Phe Ile Gln Leu Val Gln Thr Tyr
180 185 190
Asn Gln Leu Phe Glu Glu Asn Pro Ile Asn Ala Ser Gly Val Asp Ala
195 200 205
Lys Ala Ile Leu Ser Ala Arg Leu Ser Lys Ser Arg Arg Leu Glu Asn
210 215 220
Leu Ile Ala Gln Leu Pro Gly Glu Lys Lys Asn Gly Leu Phe Gly Asn
225 230 235 240
Leu Ile Ala Leu Ser Leu Gly Leu Thr Pro Asn Phe Lys Ser Asn Phe
245 250 255
Asp Leu Ala Glu Asp Ala Lys Leu Gln Leu Ser Lys Asp Thr Tyr Asp
260 265 270
Asp Asp Leu Asp Asn Leu Leu Ala Gln Ile Gly Asp Gln Tyr Ala Asp
275 280 285
Leu Phe Leu Ala Ala Lys Asn Leu Ser Asp Ala Ile Leu Leu Ser Asp
290 295 300
Ile Leu Arg Val Asn Thr Glu Ile Thr Lys Ala Pro Leu Ser Ala Ser
305 310 315 320
Met Ile Lys Arg Tyr Asp Glu His His Gln Asp Leu Thr Leu Leu Lys
325 330 335
Ala Leu Val Arg Gln Gln Leu Pro Glu Lys Tyr Lys Glu Ile Phe Phe
340 345 350
Asp Gln Ser Lys Asn Gly Tyr Ala Gly Tyr Ile Asp Gly Gly Ala Ser
355 360 365
Gln Glu Glu Phe Tyr Lys Phe Ile Lys Pro Ile Leu Glu Lys Met Asp
370 375 380
Gly Thr Glu Glu Leu Leu Val Lys Leu Asn Arg Glu Asp Leu Leu Arg
385 390 395 400
Lys Gln Arg Thr Phe Asp Asn Gly Ser Ile Pro His Gln Ile His Leu
405 410 415
Gly Glu Leu His Ala Ile Leu Arg Arg Gln Glu Asp Phe Tyr Pro Phe
420 425 430
Leu Lys Asp Asn Arg Glu Lys Ile Glu Lys Ile Leu Thr Phe Arg Ile
435 440 445
Pro Tyr Tyr Val Gly Pro Leu Ala Arg Gly Asn Ser Arg Phe Ala Trp
450 455 460
Met Thr Arg Lys Ser Glu Glu Thr Ile Thr Pro Trp Asn Phe Glu Glu
465 470 475 480
Val Val Asp Lys Gly Ala Ser Ala Gln Ser Phe Ile Glu Arg Met Thr
485 490 495
Asn Phe Asp Lys Asn Leu Pro Asn Glu Lys Val Leu Pro Lys His Ser
500 505 510
Leu Leu Tyr Glu Tyr Phe Thr Val Tyr Asn Glu Leu Thr Lys Val Lys
515 520 525
Tyr Val Thr Glu Gly Met Arg Lys Pro Ala Phe Leu Ser Gly Glu Gln
530 535 540
Lys Lys Ala Ile Val Asp Leu Leu Phe Lys Thr Asn Arg Lys Val Thr
545 550 555 560
Val Lys Gln Leu Lys Glu Asp Tyr Phe Lys Lys Ile Glu Cys Phe Asp
565 570 575
Ser Val Glu Ile Ser Gly Val Glu Asp Arg Phe Asn Ala Ser Leu Gly
580 585 590
Thr Tyr His Asp Leu Leu Lys Ile Ile Lys Asp Lys Asp Phe Leu Asp
595 600 605
Asn Glu Glu Asn Glu Asp Ile Leu Glu Asp Ile Val Leu Thr Leu Thr
610 615 620
Leu Phe Glu Asp Arg Glu Met Ile Glu Glu Arg Leu Lys Thr Tyr Ala
625 630 635 640
His Leu Phe Asp Asp Lys Val Met Lys Gln Leu Lys Arg Arg Arg Tyr
645 650 655
Thr Gly Trp Gly Arg Leu Ser Arg Lys Leu Ile Asn Gly Ile Arg Asp
660 665 670
Lys Gln Ser Gly Lys Thr Ile Leu Asp Phe Leu Lys Ser Asp Gly Phe
675 680 685
Ala Asn Arg Asn Phe Met Gln Leu Ile His Asp Asp Ser Leu Thr Phe
690 695 700
Lys Glu Asp Ile Gln Lys Ala Gln Val Ser Gly Gln Gly Asp Ser Leu
705 710 715 720
His Glu His Ile Ala Asn Leu Ala Gly Ser Pro Ala Ile Lys Lys Gly
725 730 735
Ile Leu Gln Thr Val Lys Val Val Asp Glu Leu Val Lys Val Met Gly
740 745 750
Arg His Lys Pro Glu Asn Ile Val Ile Glu Met Ala Arg Glu Asn Gln
755 760 765
Thr Thr Gln Lys Gly Gln Lys Asn Ser Arg Glu Arg Met Lys Arg Ile
770 775 780
Glu Glu Gly Ile Lys Glu Leu Gly Ser Gln Ile Leu Lys Glu His Pro
785 790 795 800
Val Glu Asn Thr Gln Leu Gln Asn Glu Lys Leu Tyr Leu Tyr Tyr Leu
805 810 815
Gln Asn Gly Arg Asp Met Tyr Val Asp Gln Glu Leu Asp Ile Asn Arg
820 825 830
Leu Ser Asp Tyr Asp Val Asp His Ile Val Pro Gln Ser Phe Leu Lys
835 840 845
Asp Asp Ser Ile Asp Asn Lys Val Leu Thr Arg Ser Asp Lys Asn Arg
850 855 860
Gly Lys Ser Asp Asn Val Pro Ser Glu Glu Val Val Lys Lys Met Lys
865 870 875 880
Asn Tyr Trp Arg Gln Leu Leu Asn Ala Lys Leu Ile Thr Gln Arg Lys
885 890 895
Phe Asp Asn Leu Thr Lys Ala Glu Arg Gly Gly Leu Ser Glu Leu Asp
900 905 910
Lys Ala Gly Phe Ile Lys Arg Gln Leu Val Glu Thr Arg Gln Ile Thr
915 920 925
Lys His Val Ala Gln Ile Leu Asp Ser Arg Met Asn Thr Lys Tyr Asp
930 935 940
Glu Asn Asp Lys Leu Ile Arg Glu Val Lys Val Ile Thr Leu Lys Ser
945 950 955 960
Lys Leu Val Ser Asp Phe Arg Lys Asp Phe Gln Phe Tyr Lys Val Arg
965 970 975
Glu Ile Asn Asn Tyr His His Ala His Asp Ala Tyr Leu Asn Ala Val
980 985 990
Val Gly Thr Ala Leu Ile Lys Lys Tyr Pro Lys Leu Glu Ser Glu Phe
995 1000 1005
Val Tyr Gly Asp Tyr Lys Val Tyr Asp Val Arg Lys Met Ile Ala Lys
1010 1015 1020
Ser Glu Gln Glu Ile Gly Lys Ala Thr Ala Lys Tyr Phe Phe Tyr Ser
1025 1030 1035 1040
Asn Ile Met Asn Phe Phe Lys Thr Glu Ile Thr Leu Ala Asn Gly Glu
1045 1050 1055
Ile Arg Lys Arg Pro Leu Ile Glu Thr Asn Gly Glu Thr Gly Glu Ile
1060 1065 1070
Val Trp Asp Lys Gly Arg Asp Phe Ala Thr Val Arg Lys Val Leu Ser
1075 1080 1085
Met Pro Gln Val Asn Ile Val Lys Lys Thr Glu Val Gln Thr Gly Gly
1090 1095 1100
Phe Ser Lys Glu Ser Ile Leu Pro Lys Arg Asn Ser Asp Lys Leu Ile
1105 1110 1115 1120
Ala Arg Lys Lys Asp Trp Asp Pro Lys Lys Tyr Gly Gly Phe Asp Ser
1125 1130 1135
Pro Thr Val Ala Tyr Ser Val Leu Val Val Ala Lys Val Glu Lys Gly
1140 1145 1150
Lys Ser Lys Lys Leu Lys Ser Val Lys Glu Leu Leu Gly Ile Thr Ile
1155 1160 1165
Met Glu Arg Ser Ser Phe Glu Lys Asn Pro Ile Asp Phe Leu Glu Ala
1170 1175 1180
Lys Gly Tyr Lys Glu Val Lys Lys Asp Leu Ile Ile Lys Leu Pro Lys
1185 1190 1195 1200
Tyr Ser Leu Phe Glu Leu Glu Asn Gly Arg Lys Arg Met Leu Ala Ser
1205 1210 1215
Ala Gly Glu Leu Gln Lys Gly Asn Glu Leu Ala Leu Pro Ser Lys Tyr
1220 1225 1230
Val Asn Phe Leu Tyr Leu Ala Ser His Tyr Glu Lys Leu Lys Gly Ser
1235 1240 1245
Pro Glu Asp Asn Glu Gln Lys Gln Leu Phe Val Glu Gln His Lys His
1250 1255 1260
Tyr Leu Asp Glu Ile Ile Glu Gln Ile Ser Glu Phe Ser Lys Arg Val
1265 1270 1275 1280
Ile Leu Ala Asp Ala Asn Leu Asp Lys Val Leu Ser Ala Tyr Asn Lys
1285 1290 1295
His Arg Asp Lys Pro Ile Arg Glu Gln Ala Glu Asn Ile Ile His Leu
1300 1305 1310
Phe Thr Leu Thr Asn Leu Gly Ala Pro Ala Ala Phe Lys Tyr Phe Asp
1315 1320 1325
Thr Thr Ile Asp Arg Lys Arg Tyr Thr Ser Thr Lys Glu Val Leu Asp
1330 1335 1340
Ala Thr Leu Ile His Gln Ser Ile Thr Gly Leu Tyr Glu Thr Arg Ile
1345 1350 1355 1360
Asp Leu Ser Gln Leu Gly Gly Asp
1365
<210> 1339
<211> 3249
<212> ДНК
<213> Искусственная последовательность
<220>
<223> N. meningitidis Cas9, кодон-оптимизированная последовательность нуклеиновой кислоты
<400> 1339
atggccgcct tcaagcccaa ccccatcaac tacatcctgg gcctggacat cggcatcgcc 60
agcgtgggct gggccatggt ggagatcgac gaggacgaga accccatctg cctgatcgac 120
ctgggtgtgc gcgtgttcga gcgcgctgag gtgcccaaga ctggtgacag tctggctatg 180
gctcgccggc ttgctcgctc tgttcggcgc cttactcgcc ggcgcgctca ccgccttctg 240
cgcgctcgcc gcctgctgaa gcgcgagggt gtgctgcagg ctgccgactt cgacgagaac 300
ggcctgatca agagcctgcc caacactcct tggcagctgc gcgctgccgc tctggaccgc 360
aagctgactc ctctggagtg gagcgccgtg ctgctgcacc tgatcaagca ccgcggctac 420
ctgagccagc gcaagaacga gggcgagacc gccgacaagg agctgggtgc tctgctgaag 480
ggcgtggccg acaacgccca cgccctgcag actggtgact tccgcactcc tgctgagctg 540
gccctgaaca agttcgagaa ggagagcggc cacatccgca accagcgcgg cgactacagc 600
cacaccttca gccgcaagga cctgcaggcc gagctgatcc tgctgttcga gaagcagaag 660
gagttcggca acccccacgt gagcggcggc ctgaaggagg gcatcgagac cctgctgatg 720
acccagcgcc ccgccctgag cggcgacgcc gtgcagaaga tgctgggcca ctgcaccttc 780
gagccagccg agcccaaggc cgccaagaac acctacaccg ccgagcgctt catctggctg 840
accaagctga acaacctgcg catcctggag cagggcagcg agcgccccct gaccgacacc 900
gagcgcgcca ccctgatgga cgagccctac cgcaagagca agctgaccta cgcccaggcc 960
cgcaagctgc tgggtctgga ggacaccgcc ttcttcaagg gcctgcgcta cggcaaggac 1020
aacgccgagg ccagcaccct gatggagatg aaggcctacc acgccatcag ccgcgccctg 1080
gagaaggagg gcctgaagga caagaagagt cctctgaacc tgagccccga gctgcaggac 1140
gagatcggca ccgccttcag cctgttcaag accgacgagg acatcaccgg ccgcctgaag 1200
gaccgcatcc agcccgagat cctggaggcc ctgctgaagc acatcagctt cgacaagttc 1260
gtgcagatca gcctgaaggc cctgcgccgc atcgtgcccc tgatggagca gggcaagcgc 1320
tacgacgagg cctgcgccga gatctacggc gaccactacg gcaagaagaa caccgaggag 1380
aagatctacc tgcctcctat ccccgccgac gagatccgca accccgtggt gctgcgcgcc 1440
ctgagccagg cccgcaaggt gatcaacggc gtggtgcgcc gctacggcag ccccgcccgc 1500
atccacatcg agaccgcccg cgaggtgggc aagagcttca aggaccgcaa ggagatcgag 1560
aagcgccagg aggagaaccg caaggaccgc gagaaggccg ccgccaagtt ccgcgagtac 1620
ttccccaact tcgtgggcga gcccaagagc aaggacatcc tgaagctgcg cctgtacgag 1680
cagcagcacg gcaagtgcct gtacagcggc aaggagatca acctgggccg cctgaacgag 1740
aagggctacg tggagatcga ccacgccctg cccttcagcc gcacctggga cgacagcttc 1800
aacaacaagg tgctggtgct gggcagcgag aaccagaaca agggcaacca gaccccctac 1860
gagtacttca acggcaagga caacagccgc gagtggcagg agttcaaggc ccgcgtggag 1920
accagccgct tcccccgcag caagaagcag cgcatcctgc tgcagaagtt cgacgaggac 1980
ggcttcaagg agcgcaacct gaacgacacc cgctacgtga accgcttcct gtgccagttc 2040
gtggccgacc gcatgcgcct gaccggcaag ggcaagaagc gcgtgttcgc cagcaacggc 2100
cagatcacca acctgctgcg cggcttctgg ggcctgcgca aggtgcgcgc cgagaacgac 2160
cgccaccacg ccctggacgc cgtggtggtg gcctgcagca ccgtggccat gcagcagaag 2220
atcacccgct tcgtgcgcta caaggagatg aacgccttcg acggtaaaac catcgacaag 2280
gagaccggcg aggtgctgca ccagaagacc cacttccccc agccctggga gttcttcgcc 2340
caggaggtga tgatccgcgt gttcggcaag cccgacggca agcccgagtt cgaggaggcc 2400
gacacccccg agaagctgcg caccctgctg gccgagaagc tgagcagccg ccctgaggcc 2460
gtgcacgagt acgtgactcc tctgttcgtg agccgcgccc ccaaccgcaa gatgagcggt 2520
cagggtcaca tggagaccgt gaagagcgcc aagcgcctgg acgagggcgt gagcgtgctg 2580
cgcgtgcccc tgacccagct gaagctgaag gacctggaga agatggtgaa ccgcgagcgc 2640
gagcccaagc tgtacgaggc cctgaaggcc cgcctggagg cccacaagga cgaccccgcc 2700
aaggccttcg ccgagccctt ctacaagtac gacaaggccg gcaaccgcac ccagcaggtg 2760
aaggccgtgc gcgtggagca ggtgcagaag accggcgtgt gggtgcgcaa ccacaacggc 2820
atcgccgaca acgccaccat ggtgcgcgtg gacgtgttcg agaagggcga caagtactac 2880
ctggtgccca tctacagctg gcaggtggcc aagggcatcc tgcccgaccg cgccgtggtg 2940
cagggcaagg acgaggagga ctggcagctg atcgacgaca gcttcaactt caagttcagc 3000
ctgcacccca acgacctggt ggaggtgatc accaagaagg cccgcatgtt cggctacttc 3060
gccagctgcc accgcggcac cggcaacatc aacatccgca tccacgacct ggaccacaag 3120
atcggcaaga acggcatcct ggagggcatc ggcgtgaaga ccgccctgag cttccagaag 3180
taccagatcg acgagctggg caaggagatc cgcccctgcc gcctgaagaa gcgccctcct 3240
gtgcgctaa 3249
<210> 1340
<211> 1082
<212> Белок
<213> Neisseria meningitidis
<400> 1340
Met Ala Ala Phe Lys Pro Asn Pro Ile Asn Tyr Ile Leu Gly Leu Asp
1 5 10 15
Ile Gly Ile Ala Ser Val Gly Trp Ala Met Val Glu Ile Asp Glu Asp
20 25 30
Glu Asn Pro Ile Cys Leu Ile Asp Leu Gly Val Arg Val Phe Glu Arg
35 40 45
Ala Glu Val Pro Lys Thr Gly Asp Ser Leu Ala Met Ala Arg Arg Leu
50 55 60
Ala Arg Ser Val Arg Arg Leu Thr Arg Arg Arg Ala His Arg Leu Leu
65 70 75 80
Arg Ala Arg Arg Leu Leu Lys Arg Glu Gly Val Leu Gln Ala Ala Asp
85 90 95
Phe Asp Glu Asn Gly Leu Ile Lys Ser Leu Pro Asn Thr Pro Trp Gln
100 105 110
Leu Arg Ala Ala Ala Leu Asp Arg Lys Leu Thr Pro Leu Glu Trp Ser
115 120 125
Ala Val Leu Leu His Leu Ile Lys His Arg Gly Tyr Leu Ser Gln Arg
130 135 140
Lys Asn Glu Gly Glu Thr Ala Asp Lys Glu Leu Gly Ala Leu Leu Lys
145 150 155 160
Gly Val Ala Asp Asn Ala His Ala Leu Gln Thr Gly Asp Phe Arg Thr
165 170 175
Pro Ala Glu Leu Ala Leu Asn Lys Phe Glu Lys Glu Ser Gly His Ile
180 185 190
Arg Asn Gln Arg Gly Asp Tyr Ser His Thr Phe Ser Arg Lys Asp Leu
195 200 205
Gln Ala Glu Leu Ile Leu Leu Phe Glu Lys Gln Lys Glu Phe Gly Asn
210 215 220
Pro His Val Ser Gly Gly Leu Lys Glu Gly Ile Glu Thr Leu Leu Met
225 230 235 240
Thr Gln Arg Pro Ala Leu Ser Gly Asp Ala Val Gln Lys Met Leu Gly
245 250 255
His Cys Thr Phe Glu Pro Ala Glu Pro Lys Ala Ala Lys Asn Thr Tyr
260 265 270
Thr Ala Glu Arg Phe Ile Trp Leu Thr Lys Leu Asn Asn Leu Arg Ile
275 280 285
Leu Glu Gln Gly Ser Glu Arg Pro Leu Thr Asp Thr Glu Arg Ala Thr
290 295 300
Leu Met Asp Glu Pro Tyr Arg Lys Ser Lys Leu Thr Tyr Ala Gln Ala
305 310 315 320
Arg Lys Leu Leu Gly Leu Glu Asp Thr Ala Phe Phe Lys Gly Leu Arg
325 330 335
Tyr Gly Lys Asp Asn Ala Glu Ala Ser Thr Leu Met Glu Met Lys Ala
340 345 350
Tyr His Ala Ile Ser Arg Ala Leu Glu Lys Glu Gly Leu Lys Asp Lys
355 360 365
Lys Ser Pro Leu Asn Leu Ser Pro Glu Leu Gln Asp Glu Ile Gly Thr
370 375 380
Ala Phe Ser Leu Phe Lys Thr Asp Glu Asp Ile Thr Gly Arg Leu Lys
385 390 395 400
Asp Arg Ile Gln Pro Glu Ile Leu Glu Ala Leu Leu Lys His Ile Ser
405 410 415
Phe Asp Lys Phe Val Gln Ile Ser Leu Lys Ala Leu Arg Arg Ile Val
420 425 430
Pro Leu Met Glu Gln Gly Lys Arg Tyr Asp Glu Ala Cys Ala Glu Ile
435 440 445
Tyr Gly Asp His Tyr Gly Lys Lys Asn Thr Glu Glu Lys Ile Tyr Leu
450 455 460
Pro Pro Ile Pro Ala Asp Glu Ile Arg Asn Pro Val Val Leu Arg Ala
465 470 475 480
Leu Ser Gln Ala Arg Lys Val Ile Asn Gly Val Val Arg Arg Tyr Gly
485 490 495
Ser Pro Ala Arg Ile His Ile Glu Thr Ala Arg Glu Val Gly Lys Ser
500 505 510
Phe Lys Asp Arg Lys Glu Ile Glu Lys Arg Gln Glu Glu Asn Arg Lys
515 520 525
Asp Arg Glu Lys Ala Ala Ala Lys Phe Arg Glu Tyr Phe Pro Asn Phe
530 535 540
Val Gly Glu Pro Lys Ser Lys Asp Ile Leu Lys Leu Arg Leu Tyr Glu
545 550 555 560
Gln Gln His Gly Lys Cys Leu Tyr Ser Gly Lys Glu Ile Asn Leu Gly
565 570 575
Arg Leu Asn Glu Lys Gly Tyr Val Glu Ile Asp His Ala Leu Pro Phe
580 585 590
Ser Arg Thr Trp Asp Asp Ser Phe Asn Asn Lys Val Leu Val Leu Gly
595 600 605
Ser Glu Asn Gln Asn Lys Gly Asn Gln Thr Pro Tyr Glu Tyr Phe Asn
610 615 620
Gly Lys Asp Asn Ser Arg Glu Trp Gln Glu Phe Lys Ala Arg Val Glu
625 630 635 640
Thr Ser Arg Phe Pro Arg Ser Lys Lys Gln Arg Ile Leu Leu Gln Lys
645 650 655
Phe Asp Glu Asp Gly Phe Lys Glu Arg Asn Leu Asn Asp Thr Arg Tyr
660 665 670
Val Asn Arg Phe Leu Cys Gln Phe Val Ala Asp Arg Met Arg Leu Thr
675 680 685
Gly Lys Gly Lys Lys Arg Val Phe Ala Ser Asn Gly Gln Ile Thr Asn
690 695 700
Leu Leu Arg Gly Phe Trp Gly Leu Arg Lys Val Arg Ala Glu Asn Asp
705 710 715 720
Arg His His Ala Leu Asp Ala Val Val Val Ala Cys Ser Thr Val Ala
725 730 735
Met Gln Gln Lys Ile Thr Arg Phe Val Arg Tyr Lys Glu Met Asn Ala
740 745 750
Phe Asp Gly Lys Thr Ile Asp Lys Glu Thr Gly Glu Val Leu His Gln
755 760 765
Lys Thr His Phe Pro Gln Pro Trp Glu Phe Phe Ala Gln Glu Val Met
770 775 780
Ile Arg Val Phe Gly Lys Pro Asp Gly Lys Pro Glu Phe Glu Glu Ala
785 790 795 800
Asp Thr Pro Glu Lys Leu Arg Thr Leu Leu Ala Glu Lys Leu Ser Ser
805 810 815
Arg Pro Glu Ala Val His Glu Tyr Val Thr Pro Leu Phe Val Ser Arg
820 825 830
Ala Pro Asn Arg Lys Met Ser Gly Gln Gly His Met Glu Thr Val Lys
835 840 845
Ser Ala Lys Arg Leu Asp Glu Gly Val Ser Val Leu Arg Val Pro Leu
850 855 860
Thr Gln Leu Lys Leu Lys Asp Leu Glu Lys Met Val Asn Arg Glu Arg
865 870 875 880
Glu Pro Lys Leu Tyr Glu Ala Leu Lys Ala Arg Leu Glu Ala His Lys
885 890 895
Asp Asp Pro Ala Lys Ala Phe Ala Glu Pro Phe Tyr Lys Tyr Asp Lys
900 905 910
Ala Gly Asn Arg Thr Gln Gln Val Lys Ala Val Arg Val Glu Gln Val
915 920 925
Gln Lys Thr Gly Val Trp Val Arg Asn His Asn Gly Ile Ala Asp Asn
930 935 940
Ala Thr Met Val Arg Val Asp Val Phe Glu Lys Gly Asp Lys Tyr Tyr
945 950 955 960
Leu Val Pro Ile Tyr Ser Trp Gln Val Ala Lys Gly Ile Leu Pro Asp
965 970 975
Arg Ala Val Val Gln Gly Lys Asp Glu Glu Asp Trp Gln Leu Ile Asp
980 985 990
Asp Ser Phe Asn Phe Lys Phe Ser Leu His Pro Asn Asp Leu Val Glu
995 1000 1005
Val Ile Thr Lys Lys Ala Arg Met Phe Gly Tyr Phe Ala Ser Cys His
1010 1015 1020
Arg Gly Thr Gly Asn Ile Asn Ile Arg Ile His Asp Leu Asp His Lys
1025 1030 1035 1040
Ile Gly Lys Asn Gly Ile Leu Glu Gly Ile Gly Val Lys Thr Ala Leu
1045 1050 1055
Ser Phe Gln Lys Tyr Gln Ile Asp Glu Leu Gly Lys Glu Ile Arg Pro
1060 1065 1070
Cys Arg Leu Lys Lys Arg Pro Pro Val Arg
1075 1080
<210> 1341
<211> 3159
<212> ДНК
<213> Искусственная последовательность
<220>
<223> S. aureus Cas9
<400> 1341
atgaaaagga actacattct ggggctggac atcgggatta caagcgtggg gtatgggatt 60
attgactatg aaacaaggga cgtgatcgac gcaggcgtca gactgttcaa ggaggccaac 120
gtggaaaaca atgagggacg gagaagcaag aggggagcca ggcgcctgaa acgacggaga 180
aggcacagaa tccagagggt gaagaaactg ctgttcgatt acaacctgct gaccgaccat 240
tctgagctga gtggaattaa tccttatgaa gccagggtga aaggcctgag tcagaagctg 300
tcagaggaag agttttccgc agctctgctg cacctggcta agcgccgagg agtgcataac 360
gtcaatgagg tggaagagga caccggcaac gagctgtcta caaaggaaca gatctcacgc 420
aatagcaaag ctctggaaga gaagtatgtc gcagagctgc agctggaacg gctgaagaaa 480
gatggcgagg tgagagggtc aattaatagg ttcaagacaa gcgactacgt caaagaagcc 540
aagcagctgc tgaaagtgca gaaggcttac caccagctgg atcagagctt catcgatact 600
tatatcgacc tgctggagac tcggagaacc tactatgagg gaccaggaga agggagcccc 660
ttcggatgga aagacatcaa ggaatggtac gagatgctga tgggacattg cacctatttt 720
ccagaagagc tgagaagcgt caagtacgct tataacgcag atctgtacaa cgccctgaat 780
gacctgaaca acctggtcat caccagggat gaaaacgaga aactggaata ctatgagaag 840
ttccagatca tcgaaaacgt gtttaagcag aagaaaaagc ctacactgaa acagattgct 900
aaggagatcc tggtcaacga agaggacatc aagggctacc gggtgacaag cactggaaaa 960
ccagagttca ccaatctgaa agtgtatcac gatattaagg acatcacagc acggaaagaa 1020
atcattgaga acgccgaact gctggatcag attgctaaga tcctgactat ctaccagagc 1080
tccgaggaca tccaggaaga gctgactaac ctgaacagcg agctgaccca ggaagagatc 1140
gaacagatta gtaatctgaa ggggtacacc ggaacacaca acctgtccct gaaagctatc 1200
aatctgattc tggatgagct gtggcataca aacgacaatc agattgcaat ctttaaccgg 1260
ctgaagctgg tcccaaaaaa ggtggacctg agtcagcaga aagagatccc aaccacactg 1320
gtggacgatt tcattctgtc acccgtggtc aagcggagct tcatccagag catcaaagtg 1380
atcaacgcca tcatcaagaa gtacggcctg cccaatgata tcattatcga gctggctagg 1440
gagaagaaca gcaaggacgc acagaagatg atcaatgaga tgcagaaacg aaaccggcag 1500
accaatgaac gcattgaaga gattatccga actaccggga aagagaacgc aaagtacctg 1560
attgaaaaaa tcaagctgca cgatatgcag gagggaaagt gtctgtattc tctggaggcc 1620
atccccctgg aggacctgct gaacaatcca ttcaactacg aggtcgatca tattatcccc 1680
agaagcgtgt ccttcgacaa ttcctttaac aacaaggtgc tggtcaagca ggaagagaac 1740
tctaaaaagg gcaataggac tcctttccag tacctgtcta gttcagattc caagatctct 1800
tacgaaacct ttaaaaagca cattctgaat ctggccaaag gaaagggccg catcagcaag 1860
accaaaaagg agtacctgct ggaagagcgg gacatcaaca gattctccgt ccagaaggat 1920
tttattaacc ggaatctggt ggacacaaga tacgctactc gcggcctgat gaatctgctg 1980
cgatcctatt tccgggtgaa caatctggat gtgaaagtca agtccatcaa cggcgggttc 2040
acatcttttc tgaggcgcaa atggaagttt aaaaaggagc gcaacaaagg gtacaagcac 2100
catgccgaag atgctctgat tatcgcaaat gccgacttca tctttaagga gtggaaaaag 2160
ctggacaaag ccaagaaagt gatggagaac cagatgttcg aagagaagca ggccgaatct 2220
atgcccgaaa tcgagacaga acaggagtac aaggagattt tcatcactcc tcaccagatc 2280
aagcatatca aggatttcaa ggactacaag tactctcacc gggtggataa aaagcccaac 2340
agagagctga tcaatgacac cctgtatagt acaagaaaag acgataaggg gaataccctg 2400
attgtgaaca atctgaacgg actgtacgac aaagataatg acaagctgaa aaagctgatc 2460
aacaaaagtc ccgagaagct gctgatgtac caccatgatc ctcagacata tcagaaactg 2520
aagctgatta tggagcagta cggcgacgag aagaacccac tgtataagta ctatgaagag 2580
actgggaact acctgaccaa gtatagcaaa aaggataatg gccccgtgat caagaagatc 2640
aagtactatg ggaacaagct gaatgcccat ctggacatca cagacgatta ccctaacagt 2700
cgcaacaagg tggtcaagct gtcactgaag ccatacagat tcgatgtcta tctggacaac 2760
ggcgtgtata aatttgtgac tgtcaagaat ctggatgtca tcaaaaagga gaactactat 2820
gaagtgaata gcaagtgcta cgaagaggct aaaaagctga aaaagattag caaccaggca 2880
gagttcatcg cctcctttta caacaacgac ctgattaaga tcaatggcga actgtatagg 2940
gtcatcgggg tgaacaatga tctgctgaac cgcattgaag tgaatatgat tgacatcact 3000
taccgagagt atctggaaaa catgaatgat aagcgccccc ctcgaattat caaaacaatt 3060
gcctctaaga ctcagagtat caaaaagtac tcaaccgaca ttctgggaaa cctgtatgag 3120
gtgaagagca aaaagcaccc tcagattatc aaaaagggc 3159
<210> 1342
<211> 1053
<212> Белок
<213> Staphylococcus aureus
<400> 1342
Met Lys Arg Asn Tyr Ile Leu Gly Leu Asp Ile Gly Ile Thr Ser Val
1 5 10 15
Gly Tyr Gly Ile Ile Asp Tyr Glu Thr Arg Asp Val Ile Asp Ala Gly
20 25 30
Val Arg Leu Phe Lys Glu Ala Asn Val Glu Asn Asn Glu Gly Arg Arg
35 40 45
Ser Lys Arg Gly Ala Arg Arg Leu Lys Arg Arg Arg Arg His Arg Ile
50 55 60
Gln Arg Val Lys Lys Leu Leu Phe Asp Tyr Asn Leu Leu Thr Asp His
65 70 75 80
Ser Glu Leu Ser Gly Ile Asn Pro Tyr Glu Ala Arg Val Lys Gly Leu
85 90 95
Ser Gln Lys Leu Ser Glu Glu Glu Phe Ser Ala Ala Leu Leu His Leu
100 105 110
Ala Lys Arg Arg Gly Val His Asn Val Asn Glu Val Glu Glu Asp Thr
115 120 125
Gly Asn Glu Leu Ser Thr Lys Glu Gln Ile Ser Arg Asn Ser Lys Ala
130 135 140
Leu Glu Glu Lys Tyr Val Ala Glu Leu Gln Leu Glu Arg Leu Lys Lys
145 150 155 160
Asp Gly Glu Val Arg Gly Ser Ile Asn Arg Phe Lys Thr Ser Asp Tyr
165 170 175
Val Lys Glu Ala Lys Gln Leu Leu Lys Val Gln Lys Ala Tyr His Gln
180 185 190
Leu Asp Gln Ser Phe Ile Asp Thr Tyr Ile Asp Leu Leu Glu Thr Arg
195 200 205
Arg Thr Tyr Tyr Glu Gly Pro Gly Glu Gly Ser Pro Phe Gly Trp Lys
210 215 220
Asp Ile Lys Glu Trp Tyr Glu Met Leu Met Gly His Cys Thr Tyr Phe
225 230 235 240
Pro Glu Glu Leu Arg Ser Val Lys Tyr Ala Tyr Asn Ala Asp Leu Tyr
245 250 255
Asn Ala Leu Asn Asp Leu Asn Asn Leu Val Ile Thr Arg Asp Glu Asn
260 265 270
Glu Lys Leu Glu Tyr Tyr Glu Lys Phe Gln Ile Ile Glu Asn Val Phe
275 280 285
Lys Gln Lys Lys Lys Pro Thr Leu Lys Gln Ile Ala Lys Glu Ile Leu
290 295 300
Val Asn Glu Glu Asp Ile Lys Gly Tyr Arg Val Thr Ser Thr Gly Lys
305 310 315 320
Pro Glu Phe Thr Asn Leu Lys Val Tyr His Asp Ile Lys Asp Ile Thr
325 330 335
Ala Arg Lys Glu Ile Ile Glu Asn Ala Glu Leu Leu Asp Gln Ile Ala
340 345 350
Lys Ile Leu Thr Ile Tyr Gln Ser Ser Glu Asp Ile Gln Glu Glu Leu
355 360 365
Thr Asn Leu Asn Ser Glu Leu Thr Gln Glu Glu Ile Glu Gln Ile Ser
370 375 380
Asn Leu Lys Gly Tyr Thr Gly Thr His Asn Leu Ser Leu Lys Ala Ile
385 390 395 400
Asn Leu Ile Leu Asp Glu Leu Trp His Thr Asn Asp Asn Gln Ile Ala
405 410 415
Ile Phe Asn Arg Leu Lys Leu Val Pro Lys Lys Val Asp Leu Ser Gln
420 425 430
Gln Lys Glu Ile Pro Thr Thr Leu Val Asp Asp Phe Ile Leu Ser Pro
435 440 445
Val Val Lys Arg Ser Phe Ile Gln Ser Ile Lys Val Ile Asn Ala Ile
450 455 460
Ile Lys Lys Tyr Gly Leu Pro Asn Asp Ile Ile Ile Glu Leu Ala Arg
465 470 475 480
Glu Lys Asn Ser Lys Asp Ala Gln Lys Met Ile Asn Glu Met Gln Lys
485 490 495
Arg Asn Arg Gln Thr Asn Glu Arg Ile Glu Glu Ile Ile Arg Thr Thr
500 505 510
Gly Lys Glu Asn Ala Lys Tyr Leu Ile Glu Lys Ile Lys Leu His Asp
515 520 525
Met Gln Glu Gly Lys Cys Leu Tyr Ser Leu Glu Ala Ile Pro Leu Glu
530 535 540
Asp Leu Leu Asn Asn Pro Phe Asn Tyr Glu Val Asp His Ile Ile Pro
545 550 555 560
Arg Ser Val Ser Phe Asp Asn Ser Phe Asn Asn Lys Val Leu Val Lys
565 570 575
Gln Glu Glu Asn Ser Lys Lys Gly Asn Arg Thr Pro Phe Gln Tyr Leu
580 585 590
Ser Ser Ser Asp Ser Lys Ile Ser Tyr Glu Thr Phe Lys Lys His Ile
595 600 605
Leu Asn Leu Ala Lys Gly Lys Gly Arg Ile Ser Lys Thr Lys Lys Glu
610 615 620
Tyr Leu Leu Glu Glu Arg Asp Ile Asn Arg Phe Ser Val Gln Lys Asp
625 630 635 640
Phe Ile Asn Arg Asn Leu Val Asp Thr Arg Tyr Ala Thr Arg Gly Leu
645 650 655
Met Asn Leu Leu Arg Ser Tyr Phe Arg Val Asn Asn Leu Asp Val Lys
660 665 670
Val Lys Ser Ile Asn Gly Gly Phe Thr Ser Phe Leu Arg Arg Lys Trp
675 680 685
Lys Phe Lys Lys Glu Arg Asn Lys Gly Tyr Lys His His Ala Glu Asp
690 695 700
Ala Leu Ile Ile Ala Asn Ala Asp Phe Ile Phe Lys Glu Trp Lys Lys
705 710 715 720
Leu Asp Lys Ala Lys Lys Val Met Glu Asn Gln Met Phe Glu Glu Lys
725 730 735
Gln Ala Glu Ser Met Pro Glu Ile Glu Thr Glu Gln Glu Tyr Lys Glu
740 745 750
Ile Phe Ile Thr Pro His Gln Ile Lys His Ile Lys Asp Phe Lys Asp
755 760 765
Tyr Lys Tyr Ser His Arg Val Asp Lys Lys Pro Asn Arg Glu Leu Ile
770 775 780
Asn Asp Thr Leu Tyr Ser Thr Arg Lys Asp Asp Lys Gly Asn Thr Leu
785 790 795 800
Ile Val Asn Asn Leu Asn Gly Leu Tyr Asp Lys Asp Asn Asp Lys Leu
805 810 815
Lys Lys Leu Ile Asn Lys Ser Pro Glu Lys Leu Leu Met Tyr His His
820 825 830
Asp Pro Gln Thr Tyr Gln Lys Leu Lys Leu Ile Met Glu Gln Tyr Gly
835 840 845
Asp Glu Lys Asn Pro Leu Tyr Lys Tyr Tyr Glu Glu Thr Gly Asn Tyr
850 855 860
Leu Thr Lys Tyr Ser Lys Lys Asp Asn Gly Pro Val Ile Lys Lys Ile
865 870 875 880
Lys Tyr Tyr Gly Asn Lys Leu Asn Ala His Leu Asp Ile Thr Asp Asp
885 890 895
Tyr Pro Asn Ser Arg Asn Lys Val Val Lys Leu Ser Leu Lys Pro Tyr
900 905 910
Arg Phe Asp Val Tyr Leu Asp Asn Gly Val Tyr Lys Phe Val Thr Val
915 920 925
Lys Asn Leu Asp Val Ile Lys Lys Glu Asn Tyr Tyr Glu Val Asn Ser
930 935 940
Lys Cys Tyr Glu Glu Ala Lys Lys Leu Lys Lys Ile Ser Asn Gln Ala
945 950 955 960
Glu Phe Ile Ala Ser Phe Tyr Asn Asn Asp Leu Ile Lys Ile Asn Gly
965 970 975
Glu Leu Tyr Arg Val Ile Gly Val Asn Asn Asp Leu Leu Asn Arg Ile
980 985 990
Glu Val Asn Met Ile Asp Ile Thr Tyr Arg Glu Tyr Leu Glu Asn Met
995 1000 1005
Asn Asp Lys Arg Pro Pro Arg Ile Ile Lys Thr Ile Ala Ser Lys Thr
1010 1015 1020
Gln Ser Ile Lys Lys Tyr Ser Thr Asp Ile Leu Gly Asn Leu Tyr Glu
1025 1030 1035 1040
Val Lys Ser Lys Lys His Pro Gln Ile Ile Lys Lys Gly
1045 1050
<210> 1343
<211> 611
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TRAC 5'-плечо гомологии
<400> 1343
ctctatcaat gagagagcaa tctcctggta atgtgataga tttcccaact taatgccaac 60
ataccataaa cctcccattc tgctaatgcc cagcctaagt tggggagacc actccagatt 120
ccaagatgta cagtttgctt tgctgggcct ttttcccatg cctgccttta ctctgccaga 180
gttatattgc tggggttttg aagaagatcc tattaaataa aagaataagc agtattatta 240
agtagccctg catttcaggt ttccttgagt ggcaggccag gcctggccgt gaacgttcac 300
tgaaatcatg gcctcttggc caagattgat agcttgtgcc tgtccctgag tcccagtcca 360
tcacgagcag ctggtttcta agatgctatt tcccgtataa agcatgagac cgtgacttgc 420
cagccccaca gagccccgcc cttgtccatc actggcatct ggactccagc ctgggttggg 480
gcaaagaggg aaatgagatc atgtcctaac cctgatcctc ttgtcccaca gatatccaga 540
accctgaccc tgccgtgtac cagctgagag actctaaatc cagtgacaag tctgtctgcc 600
tattcaccga t 611
<210> 1344
<211> 628
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TRAC 3'-плечо гомологии
<400> 1344
tttgattctc aaacaaatgt gtcacaaagt aaggattctg atgtgtatat cacagacaaa 60
actgtgctag acatgaggtc tatggacttc aagagcaaca gtgctgtggc ctggagcaac 120
aaatctgact ttgcatgtgc aaacgccttc aacaacagca ttattccaga agacaccttc 180
ttccccagcc caggtaaggg cagctttggt gccttcgcag gctgtttcct tgcttcagga 240
atggccaggt tctgcccaga gctctggtca atgatgtcta aaactcctct gattggtggt 300
ctcggcctta tccattgcca ccaaaaccct ctttttacta agaaacagtg agccttgttc 360
tggcagtcca gagaatgaca cgggaaaaaa gcagatgaag agaaggtggc aggagagggc 420
acgtggccca gcctcagtct ctccaactga gttcctgcct gcctgccttt gctcagactg 480
tttgcccctt actgctcttc taggcctcat tctaagcccc ttctccaagt tgcctctcct 540
tatttctccc tgtctgccaa aaaatctttc ccagctcact aagtcagtct cacgcagtca 600
ctcattaacc caccaatcac tgattgtg 628
<210> 1345
<211> 544
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Ef1альфа промотор с энхансером HTLV1
<400> 1345
ggatctgcga tcgctccggt gcccgtcagt gggcagagcg cacatcgccc acagtccccg 60
agaagttggg gggaggggtc ggcaattgaa ccggtgccta gagaaggtgg cgcggggtaa 120
actgggaaag tgatgtcgtg tactggctcc gcctttttcc cgagggtggg ggagaaccgt 180
atataagtgc agtagtcgcc gtgaacgttc tttttcgcaa cgggtttgcc gccagaacac 240
agctgaagct tcgaggggct cgcatctctc cttcacgcgc ccgccgccct acctgaggcc 300
gccatccacg ccggttgagt cgcgttctgc cgcctcccgc ctgtggtgcc tcctgaactg 360
cgtccgccgt ctaggtaagt ttaaagctca ggtcgagacc gggcctttgt ccggcgctcc 420
cttggagcct acctagactc agccggctct ccacgctttg cctgaccctg cttgctcaac 480
tctacgtctt tgtttcgttt tctgttctgc gccgttacag atccaagctg tgaccggcgc 540
ctac 544
<210> 1346
<211> 66
<212> ДНК
<213> Искусственная последовательность
<220>
<223> P2A нуклеотидная последовательность
<400> 1346
ggatctggag cgacgaattt tagtctactg aaacaagcgg gagacgtgga ggaaaaccct 60
ggacct 66
<210> 1347
<211> 347
<212> ДНК
<213> Искусственная последовательность
<220>
<223> MND промотор
<400> 1347
gaacagagaa acaggagaat atgggccaaa caggatatct gtggtaagca gttcctgccc 60
cggctcaggg ccaagaacag ttggaacagc agaatatggg ccaaacagga tatctgtggt 120
aagcagttcc tgccccggct cagggccaag aacagatggt ccccagatgc ggtcccgccc 180
tcagcagttt ctagagaacc atcagatgtt tccagggtgc cccaaggacc tgaaatgacc 240
ctgtgcctta tttgaactaa ccaatcagtt cgcttctcgc ttctgttcgc gcgcttctgc 300
tccccgagct ctatataagc agagctcgtt tagtgaaccg tcagatc 347
<210> 1348
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> последовательность-мишень
<400> 1348
gagaatcaaa atcggtgaat 20
<210> 1349
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> последовательность-мишень
<400> 1349
ggcctcggcg ctgacgatct 20
<210> 1350
<211> 149
<212> ДНК
<213> Искусственная последовательность
<220>
<223> иллюстративная гРНК
<400> 1350
agcgctctcg tacagagttg gcattataat acgactcact ataggggaga atcaaaatcg 60
gtgaatgttt tagagctaga aatagcaagt taaaataagg ctagtccgtt atcaacttga 120
aaaagtggca ccgagtcggt gcttttttt 149
<210> 1351
<211> 100
<212> РНК
<213> Искусственная последовательность
<220>
<223> иллюстративная гРНК
<400> 1351
gagaaucaaa aucggugaau guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcuuuu 100
<210> 1352
<211> 142
<212> Белок
<213> Homo sapiens
<220>
<223> Человеческий TCR альфа, константная область
<300>
<308> Uniprot P01848
<309> 1986-07-21
<400> 1352
Pro Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser
1 5 10 15
Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln
20 25 30
Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys
35 40 45
Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val
50 55 60
Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn
65 70 75 80
Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys
85 90 95
Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn
100 105 110
Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val
115 120 125
Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
130 135 140
<210> 1353
<211> 177
<212> Белок
<213> Homo sapiens
<220>
<223> Человеческий TCR бета, константная область 1
<300>
<308> Uniprot P01850
<309> 1986-07-21
<400> 1353
Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val Phe Glu Pro
1 5 10 15
Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys Leu
20 25 30
Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp Trp Val Asn
35 40 45
Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro Leu Lys
50 55 60
Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg Leu
65 70 75 80
Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg Cys
85 90 95
Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln Asp
100 105 110
Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly Arg
115 120 125
Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly Val Leu Ser
130 135 140
Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr Ala
145 150 155 160
Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys Asp
165 170 175
Phe
<210> 1354
<211> 178
<212> Белок
<213> Homo sapiens
<220>
<223> Человеческий TCR бета, константная область 2
<300>
<308> Uniprot A0A5B9
<309> 2010-04-20
<400> 1354
Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu Pro Ser
1 5 10 15
Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys Leu Ala
20 25 30
Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val Asn Gly
35 40 45
Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro Leu Lys Glu
50 55 60
Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg Leu Arg
65 70 75 80
Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg Cys Gln
85 90 95
Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln Asp Arg
100 105 110
Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly Arg Ala
115 120 125
Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu Ser Ala
130 135 140
Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr Ala Val
145 150 155 160
Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys Asp Ser
165 170 175
Arg Gly
<210> 1355
<211> 141
<212> Белок
<213> Homo sapiens
<220>
<223> Человеческий TCRальфа, константная область
<300>
<308> Genbank CAA26636.1
<309> 2016-07-25
<400> 1355
Asn Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg Asp Ser Lys
1 5 10 15
Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp Ser Gln Thr
20 25 30
Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr Asp Lys Thr
35 40 45
Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser Ala Val Ala
50 55 60
Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe Asn Asn Ser
65 70 75 80
Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser Ser Cys Asp
85 90 95
Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn Leu Asn Phe
100 105 110
Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu Lys Val Ala
115 120 125
Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
130 135 140
<210> 1356
<211> 179
<212> Белок
<213> Homo sapiens
<220>
<223> Человеческий TCR бета, константная область
<300>
<308> Uniprot A0A0G2JNG9
<309> 2017-06-07
<400> 1356
Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu Pro
1 5 10 15
Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys Leu
20 25 30
Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val Asn
35 40 45
Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro Leu Lys
50 55 60
Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg Leu
65 70 75 80
Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg Cys
85 90 95
Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln Asp
100 105 110
Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly Arg
115 120 125
Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu Ser
130 135 140
Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr Ala
145 150 155 160
Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys Asp
165 170 175
Ser Arg Gly
<210> 1357
<211> 25
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Иллюстративная последовательность акцепторного сайта сплайсинга
<400> 1357
ctgacctctt ctcttcctcc cacag 25
<210> 1358
<211> 13
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Иллюстративная последовательность акцепторного сайта сплайсинга
<400> 1358
tttctctcca cag 13
<210> 1359
<211> 1189
<212> ДНК
<213> Искусственная последовательность
<220>
<223> EF1альфа прмотор
<300>
<308> GenBank: J04617.1
<309> 1994-11-07
<400> 1359
cgtgaggctc cggtgcccgt cagtgggcag agcgcacatc gcccacagtc cccgagaagt 60
tggggggagg ggtcggcaat tgaaccggtg cctagagaag gtggcgcggg gtaaactggg 120
aaagtgatgt cgtgtactgg ctccgccttt ttcccgaggg tgggggagaa ccgtatataa 180
gtgcagtagt cgccgtgaac gttctttttc gcaacgggtt tgccgccaga acacaggtaa 240
gtgccgtgtg tggttcccgc gggcctggcc tctttacggg ttatggccct tgcgtgcctt 300
gaattacttc cacgcccctg gctgcagtac gtgattcttg atcccgagct tcgggttgga 360
agtgggtggg agagttcgag gccttgcgct taaggagccc cttcgcctcg tgcttgagtt 420
gaggcctggc ctgggcgctg gggccgccgc gtgcgaatct ggtggcacct tcgcgcctgt 480
ctcgctgctt tcgataagtc tctagccatt taaaattttt gatgacctgc tgcgacgctt 540
tttttctggc aagatagtct tgtaaatgcg ggccaagatc tgcacactgg tatttcggtt 600
tttggggccg cgggcggcga cggggcccgt gcgtcccagc gcacatgttc ggcgaggcgg 660
ggcctgcgag cgcggccacc gagaatcgga cgggggtagt ctcaagctgg ccggcctgct 720
ctggtgcctg gcctcgcgcc gccgtgtatc gccccgccct gggcggcaag gctggcccgg 780
tcggcaccag ttgcgtgagc ggaaagatgg ccgcttcccg gccctgctgc agggagctca 840
aaatggagga cgcggcgctc gggagagcgg gcgggtgagt cacccacaca aaggaaaagg 900
gcctttccgt cctcagccgt cgcttcatgt gactccacgg agtaccgggc gccgtccagg 960
cacctcgatt agttctcgag cttttggagt acgtcgtctt taggttgggg ggaggggttt 1020
tatgcgatgg agtttcccca cactgagtgg gtggagactg aagttaggcc agcttggcac 1080
ttgatgtaat tctccttgga atttgccctt tttgagtttg gatcttggtt cattctcaag 1140
cctcagacag tggttcaaag tttttttctt ccatttcagg tgtcgtgaa 1189
<210> 1360
<211> 1205
<212> ДНК
<213> Искусственная последовательность
<220>
<223> EF1альфа промотор
<400> 1360
cgtgaggctc cggtgcccgt cagtgggcag agcgcacatc gcccacagtc cccgagaagt 60
tggggggagg ggtcggcaat tgaaccggtg cctagagaag gtggcgcggg gtaaactggg 120
aaagtgatgt cgtgtactgg ctccgccttt ttcccgaggg tgggggagaa ccgtatataa 180
gtgcactagt cgccgtgaac gttctttttc gcaacgggtt tgccgccaga acacaggtaa 240
gtgccgtgtg tggttcccgc gggcctggcc tctttacggg ttatggccct tgcgtgcctt 300
gaattacttc cacctggctg cagtacgtga ttcttgatcc cgagcttcgg gttggaagtg 360
ggtgggagag ttcgtggcct tgcgcttaag gagccccttc gcctcgtgct tgagttgtgg 420
cctggcctgg gcgctggggc cgccgcgtgc gaatctggtg gcaccttcgc gcctgtctcg 480
ctgctttcga taagtctcta gccatttaaa atttttgatg acctgctgcg acgctttttt 540
tctggcaaga tagtcttgta aatgcgggcc aagatcagca cactggtatt tcggtttttg 600
gggccgcggg cggcgacggg gcccgtgcgt cccagcgcac atgttcggcg aggcggggcc 660
tgcgagcgcg gccaccgaga atcggacggg ggtagtctca agctgcccgg cctgctctgg 720
tgcctggcct cgcgccgccg tgtatcgccc cgccctgggc ggcaaggctg gcccggtcgg 780
caccagttgc gtgagcggaa agatggccgc ttcccggccc tgctgcaggg agcacaaaat 840
ggaggacgcg gcgctcggga gagcgggcgg gtgagtcacc cacacaaagg aaaagggcct 900
ttccgtcctc agccgtcgct tcatgtgact ccacggagta ccgggcgccg tccaggcacc 960
tcgattagtt ctccagcttt tggagtacgt cgtctttagg ttggggggag gggttttatg 1020
cgatggagtt tccccacact gagtgggtgg agactgaagt taggccagct tggcacttga 1080
tgtaattctc cttggaattt gccctttttg agtttggatc ttggttcatt ctcaagcctc 1140
agacagtggt tcaaagtttt tttcttccat ttcaggtgtc gtgaaaacta cccctaaaag 1200
ccaaa 1205
<210> 1361
<211> 181
<212> ДНК
<213> Искусственная последовательность
<220>
<223> MND промотор
<400> 1361
gggtctctct ggttagacca gatctgagcc tgggagctct ctggctaact agggaaccca 60
ctgcttaagc ctcaataaag cttgccttga gtgcttcaag tagtgtgtgc ccgtctgttg 120
tgtgactctg gtaactagag atccctcaga cccttttagt cagtgtggaa aatctctagc 180
a 181
<210> 1362
<211> 137
<212> Белок
<213> Искусственная последовательность
<220>
<223> альфа, Константная область мыши
<400> 1362
Asn Ile Gln Asn Pro Glu Pro Ala Val Tyr Gln Leu Lys Asp Pro Arg
1 5 10 15
Ser Gln Asp Ser Thr Leu Cys Leu Phe Thr Asp Phe Asp Ser Gln Ile
20 25 30
Asn Val Pro Lys Thr Met Glu Ser Gly Thr Phe Ile Thr Asp Lys Cys
35 40 45
Val Leu Asp Met Lys Ala Met Asp Ser Lys Ser Asn Gly Ala Ile Ala
50 55 60
Trp Ser Asn Gln Thr Ser Phe Thr Cys Gln Asp Ile Phe Lys Glu Thr
65 70 75 80
Asn Ala Thr Tyr Pro Ser Ser Asp Val Pro Cys Asp Ala Thr Leu Thr
85 90 95
Glu Lys Ser Phe Glu Thr Asp Met Asn Leu Asn Phe Gln Asn Leu Leu
100 105 110
Val Ile Val Leu Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu
115 120 125
Leu Met Thr Leu Arg Leu Trp Ser Ser
130 135
<210> 1363
<211> 177
<212> Белок
<213> Искусственная последовательность
<220>
<223> бета, константная область человека
<400> 1363
Glu Asp Leu Asn Lys Val Phe Pro Pro Glu Val Ala Val Phe Glu Pro
1 5 10 15
Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys Leu
20 25 30
Ala Thr Gly Phe Phe Pro Asp His Val Glu Leu Ser Trp Trp Val Asn
35 40 45
Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro Leu Lys
50 55 60
Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg Leu
65 70 75 80
Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg Cys
85 90 95
Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln Asp
100 105 110
Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly Arg
115 120 125
Ala Asp Cys Gly Phe Thr Ser Val Ser Tyr Gln Gln Gly Val Leu Ser
130 135 140
Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr Ala
145 150 155 160
Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys Asp
165 170 175
Phe
<210> 1364
<211> 138
<212> ДНК
<213> Искусственная последовательность
<220>
<223> последовательность экзона 1 константной области неполного рекомбинантного TCR альфа
<400> 1364
cagaacccag atcccgcggt atatcaactg cgcgactcaa aatcatccga taagagtgtc 60
tgtttgttta ctgacttcga cagtcaaact aatgtctctc agagcaaaga ttccgatgtc 120
tacatcactg acaagtgc 138
<210> 1365
<211> 18
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR альфа, общий консенсус CDR3
<220>
<221> ВАРИАНТ
<222> (1)...(1)
<223> X=A, I или V
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X = M, L, V, E или A
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X= R, L, N или S
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X = E, V, P, T, F, I, R или A
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X = G, I, L, A, P, R, D или H
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X= R, T, G, S, N или H
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X= G, R, A, N или отсутствует
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X= T, G или отсутствует
<220>
<221> ВАРИАНТ
<222> (9)...(9)
<223> X= отсутствует или A или G
<220>
<221> ВАРИАНТ
<222> (10)...(10)
<223> X= отсутствует или G
<220>
<221> ВАРИАНТ
<222> (11)...(11)
<223> X= отсутствует или G
<220>
<221> ВАРИАНТ
<222> (12)...(12)
<223> X= отсутствует или T;
<220>
<221> ВАРИАНТ
<222> (13)...(13)
<223> X = F, Y, A, S или отсутствует;
<220>
<221> ВАРИАНТ
<222> (14)...(14)
<223> X = G, Y или N;
<220>
<221> ВАРИАНТ
<222> (15)...(15)
<223> X = F, G, T, N, Q или Y;
<220>
<221> ВАРИАНТ
<222> (16)...(16)
<223> X = K, P, V, N или A;
<220>
<221> ВАРИАНТ
<222> (17)...(87)
<223> X17= T, L или F;
X18= I, V, T, H или N
<400> 1365
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5 10 15
Xaa Xaa
<210> 1366
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR бета, общий консенсус CDR3
<220>
<221> ВАРИАНТ
<222> (1)...(1)
<223> X = A или S
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X = S, I или V
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X = S, T или V;
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X = H, P, L, Y, T, D или Q
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X = L, G, W, F, S или R
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X = A, G, L, S или T
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X = G, E, A, T, R или отсутствует
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X = отсутствует или G
<220>
<221> ВАРИАНТ
<222> (9)...(9)
<223> X = отсутствует или G
<220>
<221> ВАРИАНТ
<222> (10)...(10)
<223> X= отсутствует, F, G, T, S или A
<220>
<221> ВАРИАНТ
<222> (11)...(11)
<223> X= T, N, H, A, S или F
<220>
<221> ВАРИАНТ
<222> (12)...(12)
<223> X = G, T, Q, D, Y или L
<220>
<221> ВАРИАНТ
<222> (13)...(13)
<223> X = E, P, T, G или W
<220>
<221> ВАРИАНТ
<222> (14)...(14)
<223> X = L, A, Q, Y или K
<220>
<221> ВАРИАНТ
<222> (15)...(15)
<223> X= F, H, Y или T
<400> 1366
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5 10 15
<210> 1367
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR альфа, общий консенсус CDR1
<220>
<221> ВАРИАНТ
<222> (1)...(1)
<223> X = T, D, N или V
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X = I или S
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X = S, D, A, P или M
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X = G, Q, P или отсутствует
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X = T, S, I или F
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X = D, Y,Q, T или S
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X = Y, G, N или Q
<400> 1367
Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5
<210> 1368
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR бета, общий консенсус CDR2
<220>
<221> ВАРИАНТ
<222> (1)...(1)
<223> X = F, Y, S или A
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X= Q, Y, V или N
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X = N, D, G, F или Q
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X= отсутствует или G
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X = E, V, N, K или S
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X = A, K, G или E
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X = Q, M, T, I или A
<400> 1368
Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5
<210> 1369
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR бета, общий консенсус CDR1
<220>
<221> ВАРИАНТ
<222> (1)...(1)
<223> X = S, M или L
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X= G, E, D, N или Q
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X = H или V
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X = V, N, E, L или T
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X = S, R, N, Y, A или M
<400> 1369
Xaa Xaa Xaa Xaa Xaa
1 5
<210> 1370
<211> 18
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR альфа E6(29-38) CDR3 консенсус
<220>
<221> ВАРИАНТ
<222> (1)...(1)
<223> X1 = A, I или V
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 = M, L или V
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 = R, L или N
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 = E, V, T, P или F
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 = G, I, L, A или P
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 = R, T, G или S
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 = G, R или отсутствует
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X8 = T, G или отсутствует
<220>
<221> ВАРИАНТ
<222> (9)...(9)
<223> X9 = отсутствует или A
<220>
<221> ВАРИАНТ
<222> (10)...(10)
<223> X10 = отсутствует или G
<220>
<221> ВАРИАНТ
<222> (11)...(11)
<223> X11 = отсутствует или G
<220>
<221> ВАРИАНТ
<222> (12)...(12)
<223> X12 = отсутствует или T
<220>
<221> ВАРИАНТ
<222> (13)...(13)
<223> X13 = отсутствует или S
<220>
<221> ВАРИАНТ
<222> (14)...(14)
<223> X14 = G, Y или N
<220>
<221> ВАРИАНТ
<222> (15)...(15)
<223> X15 = F, G или T
<220>
<221> ВАРИАНТ
<222> (16)...(16)
<223> X16 = K или P
<220>
<221> ВАРИАНТ
<222> (17)...(18)
<223> X17 = T или L;
X18 = I, V или T
<400> 1370
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5 10 15
Xaa Xaa
<210> 1371
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR альфа E6(29-38) CDR1 консенсус
<220>
<221> ВАРИАНТ
<222> (1)...(1)
<223> X1 = T, D или N
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 = I или S
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 = S, D или A
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 = G, Q, P или отсутствует
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 = T, S или I
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 = D, Y или Q
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 = Y, G, N или Q
<400> 1371
Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5
<210> 1372
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR альфа E6(29-38) CDR2 консенсус
<220>
<221> ВАРИАНТ
<222> (1)...(1)
<223> X1 = G, Q, I или V
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 = L, S, Q или Y
<220>
<221> ВАРИАНТ
<222> (3)...(3)
<223> X3 = T, G или S
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 = Y, S или отсутствует
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 = отсутствует или D
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 = отсутствует, E, Q или S
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 = S, Q, R или G
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X8 = N или E
<400> 1372
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5
<210> 1373
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR бета E6(29-38) CDR3 консенсус
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 = H, P, L или Y
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 = L, G, W, F или S
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 = A, G или L
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 = G, E, A, T или отсутствует
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X8 = F, G, T или S
<220>
<221> ВАРИАНТ
<222> (9)...(9)
<223> X9 = T, N, H или A
<220>
<221> ВАРИАНТ
<222> (10)...(10)
<223> X10 = G, T, Q, D или Y
<220>
<221> ВАРИАНТ
<222> (11)...(11)
<223> X11 = E, P, T или G
<220>
<221> ВАРИАНТ
<222> (12)...(12)
<223> X12 = L, A, Q или Y
<220>
<221> ВАРИАНТ
<222> (13)...(13)
<223> X13 = F, H, Y или T
<400> 1373
Ala Ser Ser Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5 10
<210> 1374
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> TCR альфа E7(11-19) CDR3 консенсус
<220>
<221> ВАРИАНТ
<222> (1)...(1)
<223> X1 = A или V
<220>
<221> ВАРИАНТ
<222> (2)...(2)
<223> X2 = E или V
<220>
<221> ВАРИАНТ
<222> (4)...(4)
<223> X4 = I или R
<220>
<221> ВАРИАНТ
<222> (5)...(5)
<223> X5 = R или D
<220>
<221> ВАРИАНТ
<222> (6)...(6)
<223> X6 = G или N
<220>
<221> ВАРИАНТ
<222> (7)...(7)
<223> X7 = F или Y
<220>
<221> ВАРИАНТ
<222> (8)...(8)
<223> X8 = N или Q
<220>
<221> ВАРИАНТ
<222> (9)...(9)
<223> X9 = V или N
<220>
<221> ВАРИАНТ
<222> (10)...(10)
<223> X10 = L или F
<220>
<221> ВАРИАНТ
<222> (11)...(11)
<223> X11 = H или V
<400> 1374
Xaa Xaa Ser Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5 10
<210> 1375
<211> 876
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 66 бета, кодон-оптимизированный/ цистеин-модифицированный
<400> 1375
gagcccgaag tgacccagac acccagccac caggtcacac agatgggcca ggaagtgatc 60
ctgcgctgcg tgcccatcag caaccacctg tacttctact ggtacagaca gatcctgggc 120
cagaaggtgg aattcctcgt cagcttctac aacaacgaga tcagcgagaa gtccgagatt 180
ttcgacgacc agttcagcgt ggaacggccc gacggcagca acttcaccct gaagatcaga 240
agcaccaagc tcgaggacag cgccatgtac ttttgcgcca ccaccggcag aagcagctac 300
gagcagtact tcggccctgg caccagactg accgtgaccg aagatctgaa gaacgtcttc 360
ccccctgagg tggcagtctt tgaaccctct gaggccgaaa tcagtcacac tcagaaagcc 420
accctggtgt gcctggctac agggttttac cctgatcacg tggagctgag ttggtgggtc 480
aacggcaagg aagtgcattc aggggtctgc actgacccac agcccctgaa agagcagcca 540
gcactgaatg attccaggta ctgtctgagt tcacggctga gagtgtctgc cacattttgg 600
cagaaccccc gaaatcattt ccggtgccag gtccagtttt atggcctgag cgagaatgac 660
gaatggacac aggatcgggc taagcccgtg actcagattg tctccgccga ggcttggggg 720
agagcagact gtggattcac ctccgaatct taccagcagg gcgtgctgtc tgccaccatc 780
ctgtacgaga ttctgctggg gaaggctaca ctgtatgccg tgctggtgag cgccctggtg 840
ctgatggcaa tggtcaagag aaaagacagc agggga 876
<210> 1376
<211> 292
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 66 - бета нативный
<400> 1376
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Thr Thr Gly
85 90 95
Arg Ser Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu
115 120 125
Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys
130 135 140
Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val
145 150 155 160
Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro Gln Pro Leu
165 170 175
Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg
180 185 190
Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg
195 200 205
Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln
210 215 220
Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly
225 230 235 240
Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu
245 250 255
Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr
260 265 270
Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys
275 280 285
Asp Ser Arg Gly
290
<210> 1377
<211> 292
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 66 - бета, цистеин-модифицированный
<400> 1377
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Thr Thr Gly
85 90 95
Arg Ser Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala Val Phe Glu
115 120 125
Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr Leu Val Cys
130 135 140
Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser Trp Trp Val
145 150 155 160
Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro Gln Pro Leu
165 170 175
Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu Ser Ser Arg
180 185 190
Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn His Phe Arg
195 200 205
Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu Trp Thr Gln
210 215 220
Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu Ala Trp Gly
225 230 235 240
Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln Gly Val Leu
245 250 255
Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala Thr Leu Tyr
260 265 270
Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val Lys Arg Lys
275 280 285
Asp Ser Arg Gly
290
<210> 1378
<211> 311
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 66 - бета нативный
<400> 1378
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Thr Thr Gly Arg Ser Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 1379
<211> 311
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 66 - бета, цистеин-модифицированный
<400> 1379
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Thr Thr Gly Arg Ser Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly
305 310
<210> 1380
<211> 113
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 66 - бета, вариабельная область
<400> 1380
Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr Gln Met Gly
1 5 10 15
Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His Leu Tyr Phe
20 25 30
Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe Leu Val Ser
35 40 45
Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe Asp Asp Gln
50 55 60
Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu Lys Ile Arg
65 70 75 80
Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala Thr Thr Gly
85 90 95
Arg Ser Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg Leu Thr Val
100 105 110
Thr
<210> 1381
<211> 11
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 66 бета CDR3
<400> 1381
Ala Thr Thr Gly Arg Ser Ser Tyr Glu Gln Tyr
1 5 10
<210> 1382
<211> 1833
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 66 кодон-оптимизированный/ цистеин-модифицированный, полноразмерная
последовательность
<400> 1382
atggatacct ggctcgtgtg ctgggccatc ttcagcctgc tgaaggccgg actgaccgag 60
cccgaagtga cccagacacc cagccaccag gtcacacaga tgggccagga agtgatcctg 120
cgctgcgtgc ccatcagcaa ccacctgtac ttctactggt acagacagat cctgggccag 180
aaggtggaat tcctcgtcag cttctacaac aacgagatca gcgagaagtc cgagattttc 240
gacgaccagt tcagcgtgga acggcccgac ggcagcaact tcaccctgaa gatcagaagc 300
accaagctcg aggacagcgc catgtacttt tgcgccacca ccggcagaag cagctacgag 360
cagtacttcg gccctggcac cagactgacc gtgaccgaag atctgaagaa cgtcttcccc 420
cctgaggtgg cagtctttga accctctgag gccgaaatca gtcacactca gaaagccacc 480
ctggtgtgcc tggctacagg gttttaccct gatcacgtgg agctgagttg gtgggtcaac 540
ggcaaggaag tgcattcagg ggtctgcact gacccacagc ccctgaaaga gcagccagca 600
ctgaatgatt ccaggtactg tctgagttca cggctgagag tgtctgccac attttggcag 660
aacccccgaa atcatttccg gtgccaggtc cagttttatg gcctgagcga gaatgacgaa 720
tggacacagg atcgggctaa gcccgtgact cagattgtct ccgccgaggc ttgggggaga 780
gcagactgtg gattcacctc cgaatcttac cagcagggcg tgctgtctgc caccatcctg 840
tacgagattc tgctggggaa ggctacactg tatgccgtgc tggtgagcgc cctggtgctg 900
atggcaatgg tcaagagaaa agacagcagg ggaggctctg gcgccaccaa ctttagcctg 960
ctgaagcagg ccggcgatgt ggaggagaat cctggcccaa tgatgatcag cctgcgggtg 1020
ctgctggtca tcctgtggct gcagctgagc tgggtctggt cccagcggaa agaggtggaa 1080
caggaccctg gccccttcaa cgtgccagag ggcgctaccg tggccttcaa ctgcacctac 1140
agcaacagcg ccagccagag cttcttctgg tacagacagg actgccggaa agaacccaag 1200
ctgctgatga gcgtgtacag cagcggcaac gaggacggca gattcaccgc ccagctgaac 1260
cgggccagcc agtacatctc cctgctgatc cgggacagca agctgagcga cagcgccacc 1320
tacctgtgcg tggtcaacag cagacctaga agcagcggcg gcagctacat ccccaccttt 1380
ggcagaggca ccagcctgat cgtgcacccc tatatccaga atcccgaccc tgccgtctac 1440
cagctgaggg actcaaagag ctccgataaa agcgtgtgcc tgttcaccga ctttgattct 1500
cagacaaacg tcagccagtc caaggacagt gacgtgtaca ttacagacaa atgcgtgctg 1560
gatatgcgct ccatggactt caagtctaac agtgcagtgg cctggtccaa caagtctgat 1620
ttcgcttgcg caaacgcctt taacaattca atcattccag aggatacctt ctttccaagc 1680
cccgaatcta gttgtgacgt gaagctggtc gagaaatctt tcgaaacaga tactaacctg 1740
aattttcaga atctgagcgt gatcggcttc cgcattctgc tgctgaaagt ggctgggttt 1800
aatctgctga tgactctgcg gctgtggtct tcc 1833
<210> 1383
<211> 611
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 66 полноразмерная последовательность, нативный
<400> 1383
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Thr Thr Gly Arg Ser Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Ser Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser Leu
305 310 315 320
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Met Ile
325 330 335
Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu Ser Trp Val
340 345 350
Trp Ser Gln Arg Lys Glu Val Glu Gln Asp Pro Gly Pro Phe Asn Val
355 360 365
Pro Glu Gly Ala Thr Val Ala Phe Asn Cys Thr Tyr Ser Asn Ser Ala
370 375 380
Ser Gln Ser Phe Phe Trp Tyr Arg Gln Asp Cys Arg Lys Glu Pro Lys
385 390 395 400
Leu Leu Met Ser Val Tyr Ser Ser Gly Asn Glu Asp Gly Arg Phe Thr
405 410 415
Ala Gln Leu Asn Arg Ala Ser Gln Tyr Ile Ser Leu Leu Ile Arg Asp
420 425 430
Ser Lys Leu Ser Asp Ser Ala Thr Tyr Leu Cys Val Val Asn Ser Arg
435 440 445
Pro Arg Ser Ser Gly Gly Ser Tyr Ile Pro Thr Phe Gly Arg Gly Thr
450 455 460
Ser Leu Ile Val His Pro Tyr Ile Gln Asn Pro Asp Pro Ala Val Tyr
465 470 475 480
Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr
485 490 495
Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val
500 505 510
Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys
515 520 525
Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala
530 535 540
Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser
545 550 555 560
Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr
565 570 575
Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile
580 585 590
Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu
595 600 605
Trp Ser Ser
610
<210> 1384
<211> 611
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 66 полноразмерная последовательность, цистеин-модифицированная
<400> 1384
Met Asp Thr Trp Leu Val Cys Trp Ala Ile Phe Ser Leu Leu Lys Ala
1 5 10 15
Gly Leu Thr Glu Pro Glu Val Thr Gln Thr Pro Ser His Gln Val Thr
20 25 30
Gln Met Gly Gln Glu Val Ile Leu Arg Cys Val Pro Ile Ser Asn His
35 40 45
Leu Tyr Phe Tyr Trp Tyr Arg Gln Ile Leu Gly Gln Lys Val Glu Phe
50 55 60
Leu Val Ser Phe Tyr Asn Asn Glu Ile Ser Glu Lys Ser Glu Ile Phe
65 70 75 80
Asp Asp Gln Phe Ser Val Glu Arg Pro Asp Gly Ser Asn Phe Thr Leu
85 90 95
Lys Ile Arg Ser Thr Lys Leu Glu Asp Ser Ala Met Tyr Phe Cys Ala
100 105 110
Thr Thr Gly Arg Ser Ser Tyr Glu Gln Tyr Phe Gly Pro Gly Thr Arg
115 120 125
Leu Thr Val Thr Glu Asp Leu Lys Asn Val Phe Pro Pro Glu Val Ala
130 135 140
Val Phe Glu Pro Ser Glu Ala Glu Ile Ser His Thr Gln Lys Ala Thr
145 150 155 160
Leu Val Cys Leu Ala Thr Gly Phe Tyr Pro Asp His Val Glu Leu Ser
165 170 175
Trp Trp Val Asn Gly Lys Glu Val His Ser Gly Val Cys Thr Asp Pro
180 185 190
Gln Pro Leu Lys Glu Gln Pro Ala Leu Asn Asp Ser Arg Tyr Cys Leu
195 200 205
Ser Ser Arg Leu Arg Val Ser Ala Thr Phe Trp Gln Asn Pro Arg Asn
210 215 220
His Phe Arg Cys Gln Val Gln Phe Tyr Gly Leu Ser Glu Asn Asp Glu
225 230 235 240
Trp Thr Gln Asp Arg Ala Lys Pro Val Thr Gln Ile Val Ser Ala Glu
245 250 255
Ala Trp Gly Arg Ala Asp Cys Gly Phe Thr Ser Glu Ser Tyr Gln Gln
260 265 270
Gly Val Leu Ser Ala Thr Ile Leu Tyr Glu Ile Leu Leu Gly Lys Ala
275 280 285
Thr Leu Tyr Ala Val Leu Val Ser Ala Leu Val Leu Met Ala Met Val
290 295 300
Lys Arg Lys Asp Ser Arg Gly Gly Ser Gly Ala Thr Asn Phe Ser Leu
305 310 315 320
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Met Ile
325 330 335
Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu Ser Trp Val
340 345 350
Trp Ser Gln Arg Lys Glu Val Glu Gln Asp Pro Gly Pro Phe Asn Val
355 360 365
Pro Glu Gly Ala Thr Val Ala Phe Asn Cys Thr Tyr Ser Asn Ser Ala
370 375 380
Ser Gln Ser Phe Phe Trp Tyr Arg Gln Asp Cys Arg Lys Glu Pro Lys
385 390 395 400
Leu Leu Met Ser Val Tyr Ser Ser Gly Asn Glu Asp Gly Arg Phe Thr
405 410 415
Ala Gln Leu Asn Arg Ala Ser Gln Tyr Ile Ser Leu Leu Ile Arg Asp
420 425 430
Ser Lys Leu Ser Asp Ser Ala Thr Tyr Leu Cys Val Val Asn Ser Arg
435 440 445
Pro Arg Ser Ser Gly Gly Ser Tyr Ile Pro Thr Phe Gly Arg Gly Thr
450 455 460
Ser Leu Ile Val His Pro Tyr Ile Gln Asn Pro Asp Pro Ala Val Tyr
465 470 475 480
Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr
485 490 495
Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val
500 505 510
Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys
515 520 525
Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala
530 535 540
Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser
545 550 555 560
Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr
565 570 575
Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile
580 585 590
Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu
595 600 605
Trp Ser Ser
610
<210> 1385
<211> 768
<212> ДНК
<213> Homo sapiens
<220>
<223> TCR 66 альфа, кодон-оптимизированный/ цистеин-модифицированный
<400> 1385
cggaaagagg tggaacagga ccctggcccc ttcaacgtgc cagagggcgc taccgtggcc 60
ttcaactgca cctacagcaa cagcgccagc cagagcttct tctggtacag acaggactgc 120
cggaaagaac ccaagctgct gatgagcgtg tacagcagcg gcaacgagga cggcagattc 180
accgcccagc tgaaccgggc cagccagtac atctccctgc tgatccggga cagcaagctg 240
agcgacagcg ccacctacct gtgcgtggtc aacagcagac ctagaagcag cggcggcagc 300
tacatcccca cctttggcag aggcaccagc ctgatcgtgc acccctatat ccagaatccc 360
gaccctgccg tctaccagct gagggactca aagagctccg ataaaagcgt gtgcctgttc 420
accgactttg attctcagac aaacgtcagc cagtccaagg acagtgacgt gtacattaca 480
gacaaatgcg tgctggatat gcgctccatg gacttcaagt ctaacagtgc agtggcctgg 540
tccaacaagt ctgatttcgc ttgcgcaaac gcctttaaca attcaatcat tccagaggat 600
accttctttc caagccccga atctagttgt gacgtgaagc tggtcgagaa atctttcgaa 660
acagatacta acctgaattt tcagaatctg agcgtgatcg gcttccgcat tctgctgctg 720
aaagtggctg ggtttaatct gctgatgact ctgcggctgt ggtcttcc 768
<210> 1386
<211> 256
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 66 - альфа нативный
<400> 1386
Arg Lys Glu Val Glu Gln Asp Pro Gly Pro Phe Asn Val Pro Glu Gly
1 5 10 15
Ala Thr Val Ala Phe Asn Cys Thr Tyr Ser Asn Ser Ala Ser Gln Ser
20 25 30
Phe Phe Trp Tyr Arg Gln Asp Cys Arg Lys Glu Pro Lys Leu Leu Met
35 40 45
Ser Val Tyr Ser Ser Gly Asn Glu Asp Gly Arg Phe Thr Ala Gln Leu
50 55 60
Asn Arg Ala Ser Gln Tyr Ile Ser Leu Leu Ile Arg Asp Ser Lys Leu
65 70 75 80
Ser Asp Ser Ala Thr Tyr Leu Cys Val Val Asn Ser Arg Pro Arg Ser
85 90 95
Ser Gly Gly Ser Tyr Ile Pro Thr Phe Gly Arg Gly Thr Ser Leu Ile
100 105 110
Val His Pro Tyr Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg
115 120 125
Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp
130 135 140
Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr
145 150 155 160
Asp Lys Thr Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser
165 170 175
Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe
180 185 190
Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser
195 200 205
Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn
210 215 220
Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu
225 230 235 240
Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250 255
<210> 1387
<211> 256
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 66 - альфа, цистеин-модифицированный
<400> 1387
Arg Lys Glu Val Glu Gln Asp Pro Gly Pro Phe Asn Val Pro Glu Gly
1 5 10 15
Ala Thr Val Ala Phe Asn Cys Thr Tyr Ser Asn Ser Ala Ser Gln Ser
20 25 30
Phe Phe Trp Tyr Arg Gln Asp Cys Arg Lys Glu Pro Lys Leu Leu Met
35 40 45
Ser Val Tyr Ser Ser Gly Asn Glu Asp Gly Arg Phe Thr Ala Gln Leu
50 55 60
Asn Arg Ala Ser Gln Tyr Ile Ser Leu Leu Ile Arg Asp Ser Lys Leu
65 70 75 80
Ser Asp Ser Ala Thr Tyr Leu Cys Val Val Asn Ser Arg Pro Arg Ser
85 90 95
Ser Gly Gly Ser Tyr Ile Pro Thr Phe Gly Arg Gly Thr Ser Leu Ile
100 105 110
Val His Pro Tyr Ile Gln Asn Pro Asp Pro Ala Val Tyr Gln Leu Arg
115 120 125
Asp Ser Lys Ser Ser Asp Lys Ser Val Cys Leu Phe Thr Asp Phe Asp
130 135 140
Ser Gln Thr Asn Val Ser Gln Ser Lys Asp Ser Asp Val Tyr Ile Thr
145 150 155 160
Asp Lys Cys Val Leu Asp Met Arg Ser Met Asp Phe Lys Ser Asn Ser
165 170 175
Ala Val Ala Trp Ser Asn Lys Ser Asp Phe Ala Cys Ala Asn Ala Phe
180 185 190
Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe Phe Pro Ser Pro Glu Ser
195 200 205
Ser Cys Asp Val Lys Leu Val Glu Lys Ser Phe Glu Thr Asp Thr Asn
210 215 220
Leu Asn Phe Gln Asn Leu Ser Val Ile Gly Phe Arg Ile Leu Leu Leu
225 230 235 240
Lys Val Ala Gly Phe Asn Leu Leu Met Thr Leu Arg Leu Trp Ser Ser
245 250 255
<210> 1388
<211> 278
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 66 - альфа нативный
<400> 1388
Met Met Ile Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu
1 5 10 15
Ser Trp Val Trp Ser Gln Arg Lys Glu Val Glu Gln Asp Pro Gly Pro
20 25 30
Phe Asn Val Pro Glu Gly Ala Thr Val Ala Phe Asn Cys Thr Tyr Ser
35 40 45
Asn Ser Ala Ser Gln Ser Phe Phe Trp Tyr Arg Gln Asp Cys Arg Lys
50 55 60
Glu Pro Lys Leu Leu Met Ser Val Tyr Ser Ser Gly Asn Glu Asp Gly
65 70 75 80
Arg Phe Thr Ala Gln Leu Asn Arg Ala Ser Gln Tyr Ile Ser Leu Leu
85 90 95
Ile Arg Asp Ser Lys Leu Ser Asp Ser Ala Thr Tyr Leu Cys Val Val
100 105 110
Asn Ser Arg Pro Arg Ser Ser Gly Gly Ser Tyr Ile Pro Thr Phe Gly
115 120 125
Arg Gly Thr Ser Leu Ile Val His Pro Tyr Ile Gln Asn Pro Asp Pro
130 135 140
Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys
145 150 155 160
Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp
165 170 175
Ser Asp Val Tyr Ile Thr Asp Lys Thr Val Leu Asp Met Arg Ser Met
180 185 190
Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe
195 200 205
Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe
210 215 220
Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser
225 230 235 240
Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly
245 250 255
Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr
260 265 270
Leu Arg Leu Trp Ser Ser
275
<210> 1389
<211> 278
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 66 - альфа, цистеин-модифицированный
<400> 1389
Met Met Ile Ser Leu Arg Val Leu Leu Val Ile Leu Trp Leu Gln Leu
1 5 10 15
Ser Trp Val Trp Ser Gln Arg Lys Glu Val Glu Gln Asp Pro Gly Pro
20 25 30
Phe Asn Val Pro Glu Gly Ala Thr Val Ala Phe Asn Cys Thr Tyr Ser
35 40 45
Asn Ser Ala Ser Gln Ser Phe Phe Trp Tyr Arg Gln Asp Cys Arg Lys
50 55 60
Glu Pro Lys Leu Leu Met Ser Val Tyr Ser Ser Gly Asn Glu Asp Gly
65 70 75 80
Arg Phe Thr Ala Gln Leu Asn Arg Ala Ser Gln Tyr Ile Ser Leu Leu
85 90 95
Ile Arg Asp Ser Lys Leu Ser Asp Ser Ala Thr Tyr Leu Cys Val Val
100 105 110
Asn Ser Arg Pro Arg Ser Ser Gly Gly Ser Tyr Ile Pro Thr Phe Gly
115 120 125
Arg Gly Thr Ser Leu Ile Val His Pro Tyr Ile Gln Asn Pro Asp Pro
130 135 140
Ala Val Tyr Gln Leu Arg Asp Ser Lys Ser Ser Asp Lys Ser Val Cys
145 150 155 160
Leu Phe Thr Asp Phe Asp Ser Gln Thr Asn Val Ser Gln Ser Lys Asp
165 170 175
Ser Asp Val Tyr Ile Thr Asp Lys Cys Val Leu Asp Met Arg Ser Met
180 185 190
Asp Phe Lys Ser Asn Ser Ala Val Ala Trp Ser Asn Lys Ser Asp Phe
195 200 205
Ala Cys Ala Asn Ala Phe Asn Asn Ser Ile Ile Pro Glu Asp Thr Phe
210 215 220
Phe Pro Ser Pro Glu Ser Ser Cys Asp Val Lys Leu Val Glu Lys Ser
225 230 235 240
Phe Glu Thr Asp Thr Asn Leu Asn Phe Gln Asn Leu Ser Val Ile Gly
245 250 255
Phe Arg Ile Leu Leu Leu Lys Val Ala Gly Phe Asn Leu Leu Met Thr
260 265 270
Leu Arg Leu Trp Ser Ser
275
<210> 1390
<211> 115
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 66- альфа, вариабельная область
<400> 1390
Arg Lys Glu Val Glu Gln Asp Pro Gly Pro Phe Asn Val Pro Glu Gly
1 5 10 15
Ala Thr Val Ala Phe Asn Cys Thr Tyr Ser Asn Ser Ala Ser Gln Ser
20 25 30
Phe Phe Trp Tyr Arg Gln Asp Cys Arg Lys Glu Pro Lys Leu Leu Met
35 40 45
Ser Val Tyr Ser Ser Gly Asn Glu Asp Gly Arg Phe Thr Ala Gln Leu
50 55 60
Asn Arg Ala Ser Gln Tyr Ile Ser Leu Leu Ile Arg Asp Ser Lys Leu
65 70 75 80
Ser Asp Ser Ala Thr Tyr Leu Cys Val Val Asn Ser Arg Pro Arg Ser
85 90 95
Ser Gly Gly Ser Tyr Ile Pro Thr Phe Gly Arg Gly Thr Ser Leu Ile
100 105 110
Val His Pro
115
<210> 1391
<211> 16
<212> Белок
<213> Homo sapiens
<220>
<223> TCR 66 - альфа CDR3
<400> 1391
Val Val Asn Ser Arg Pro Arg Ser Ser Gly Gly Ser Tyr Ile Pro Thr
1 5 10 15
<---
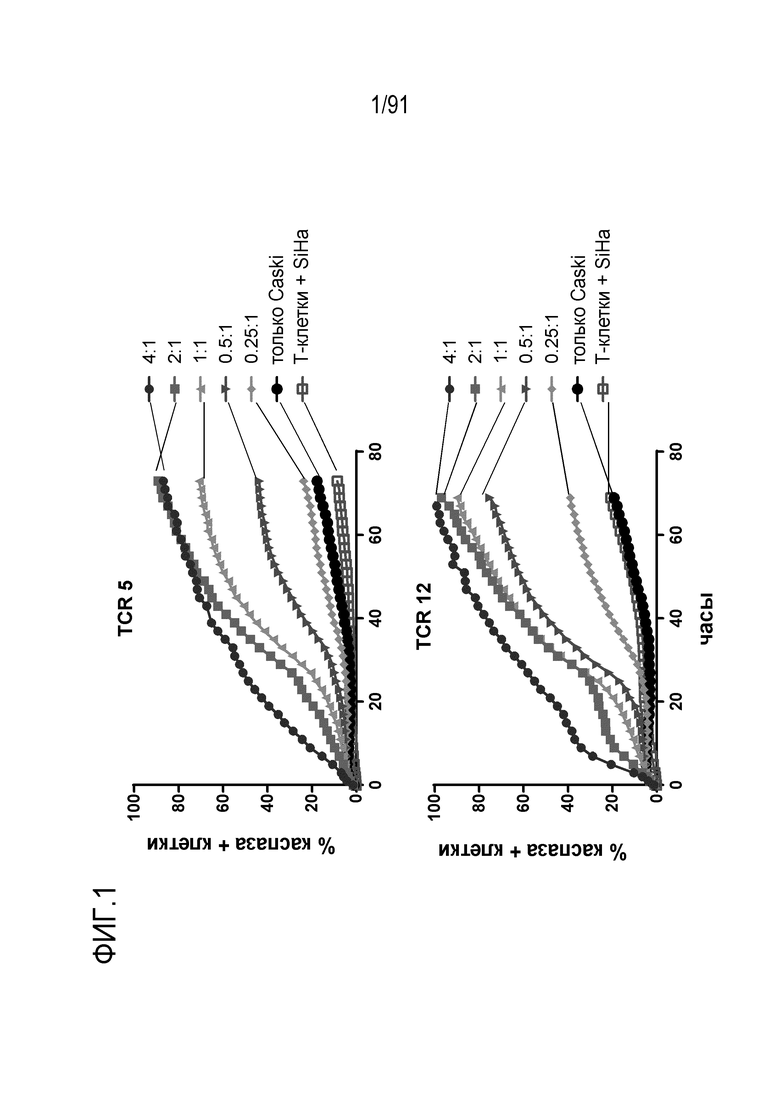
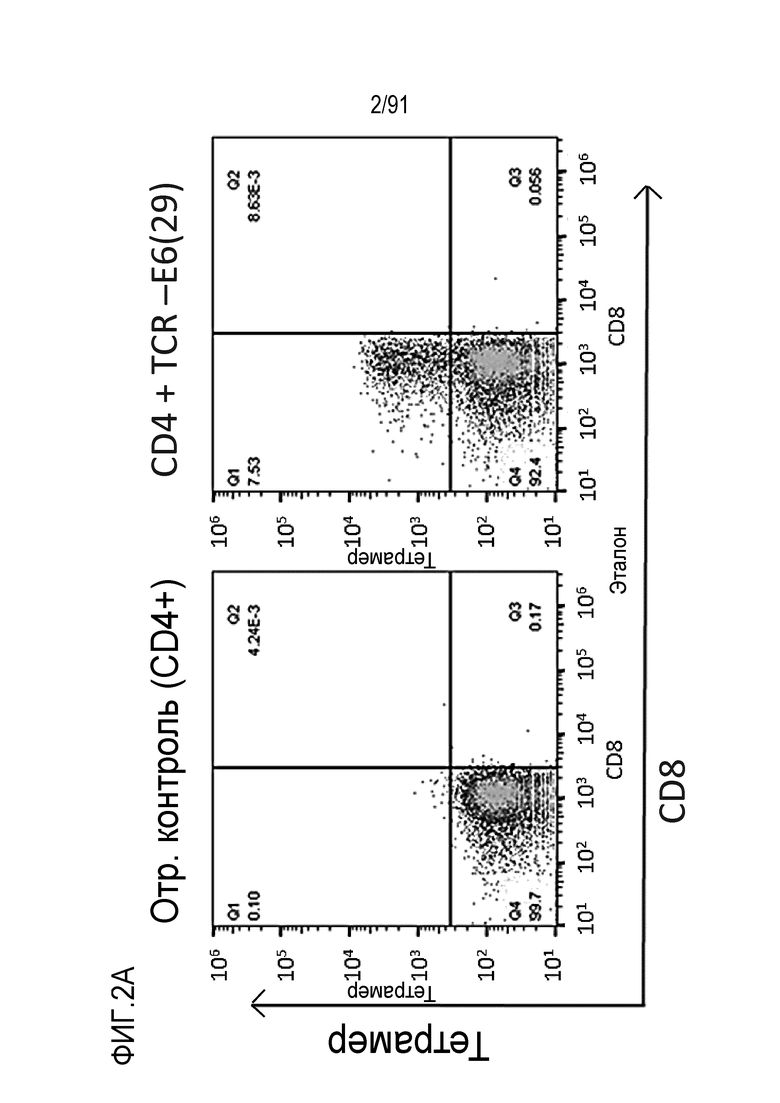
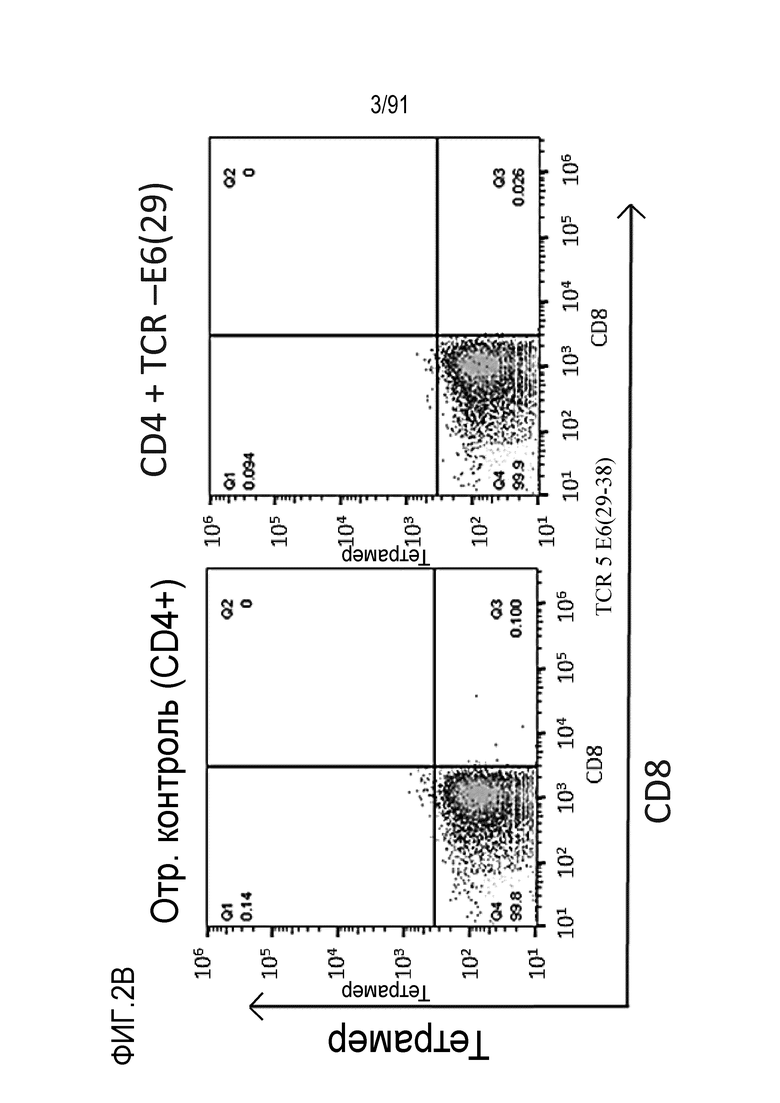
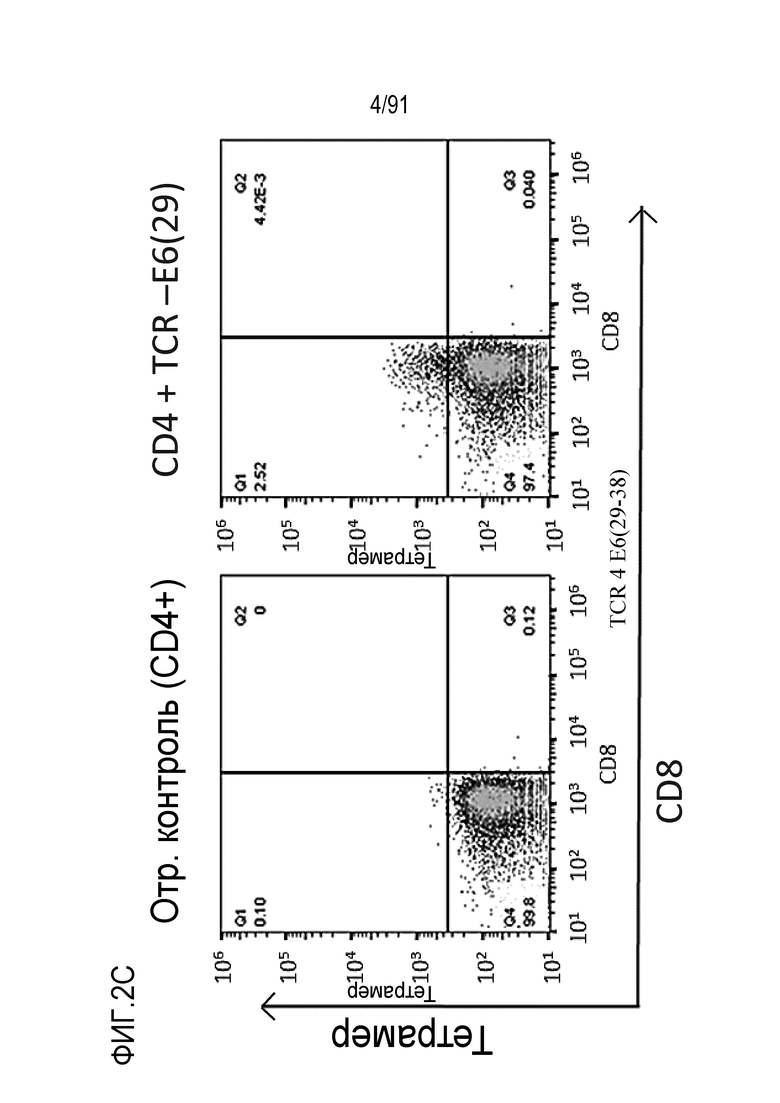
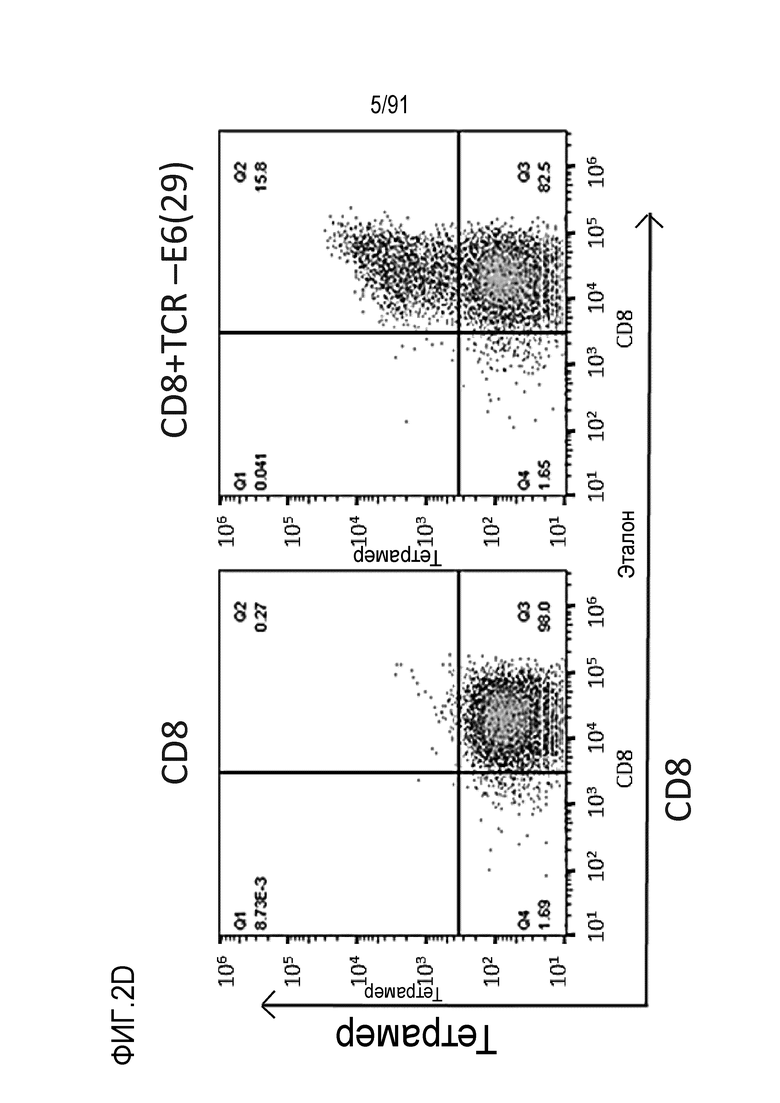
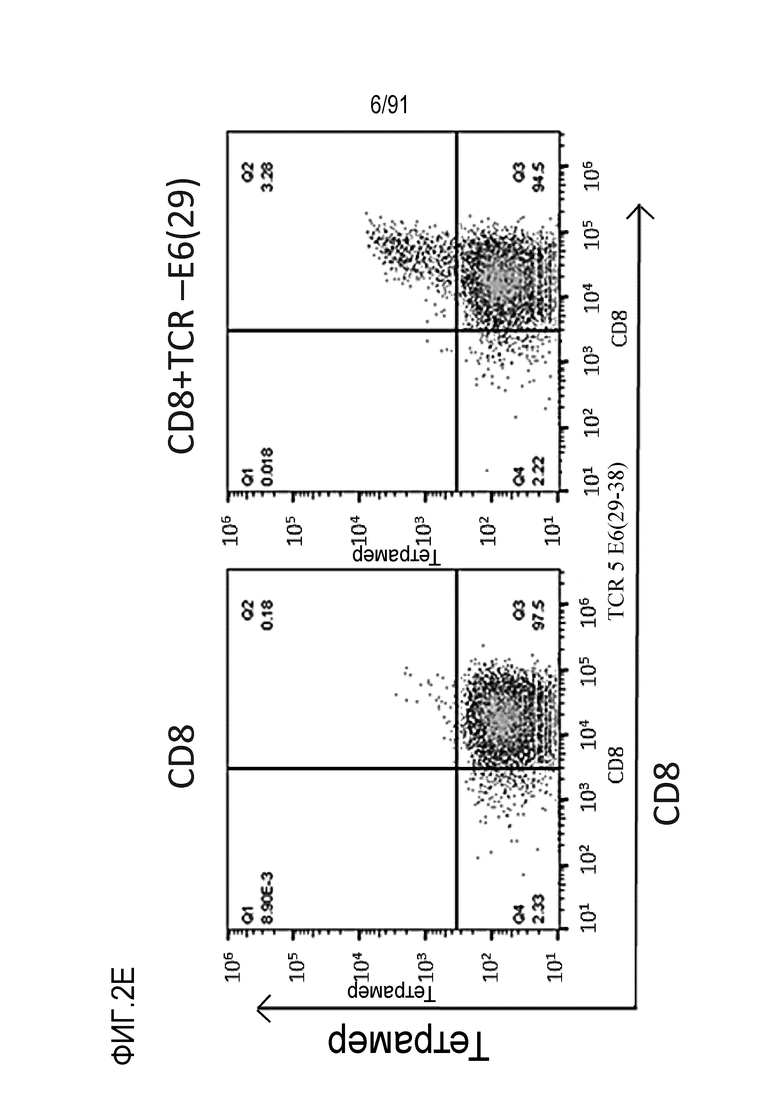
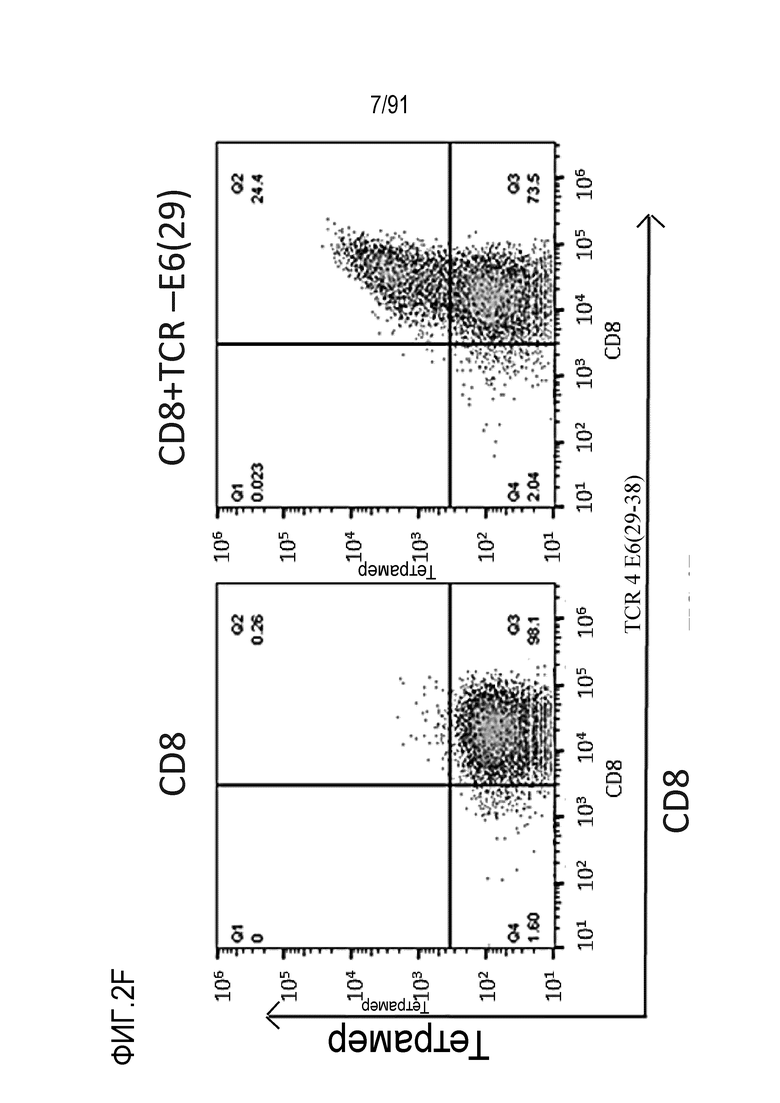

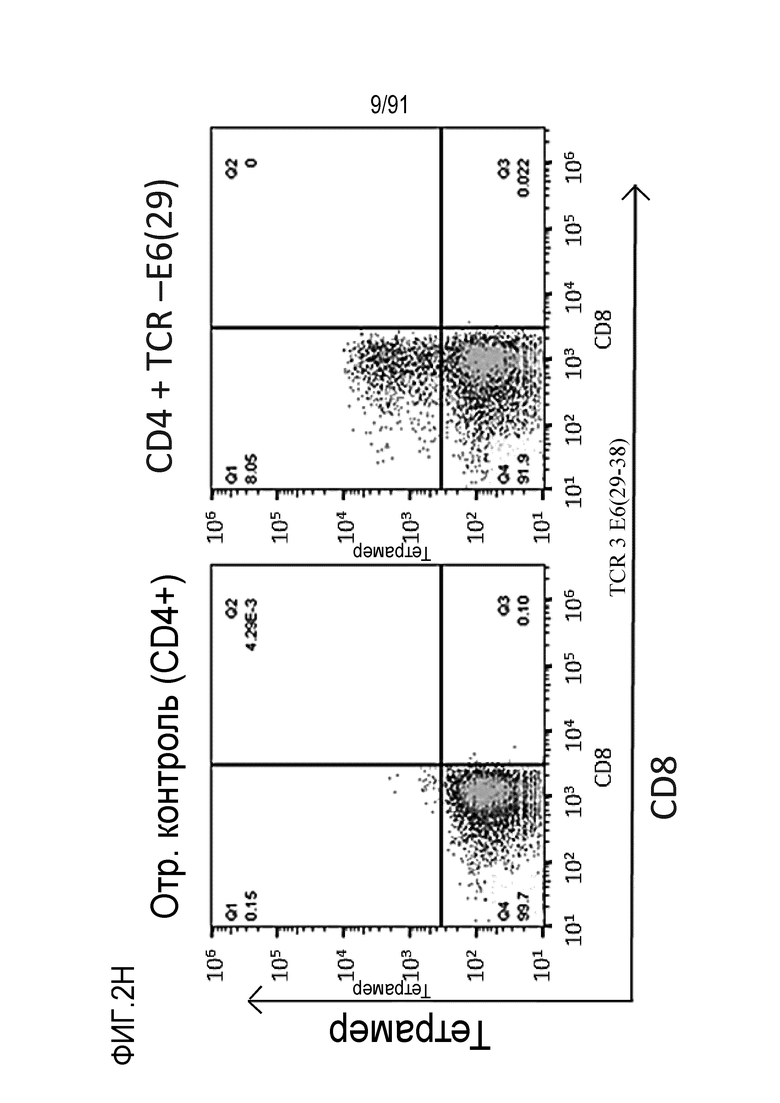
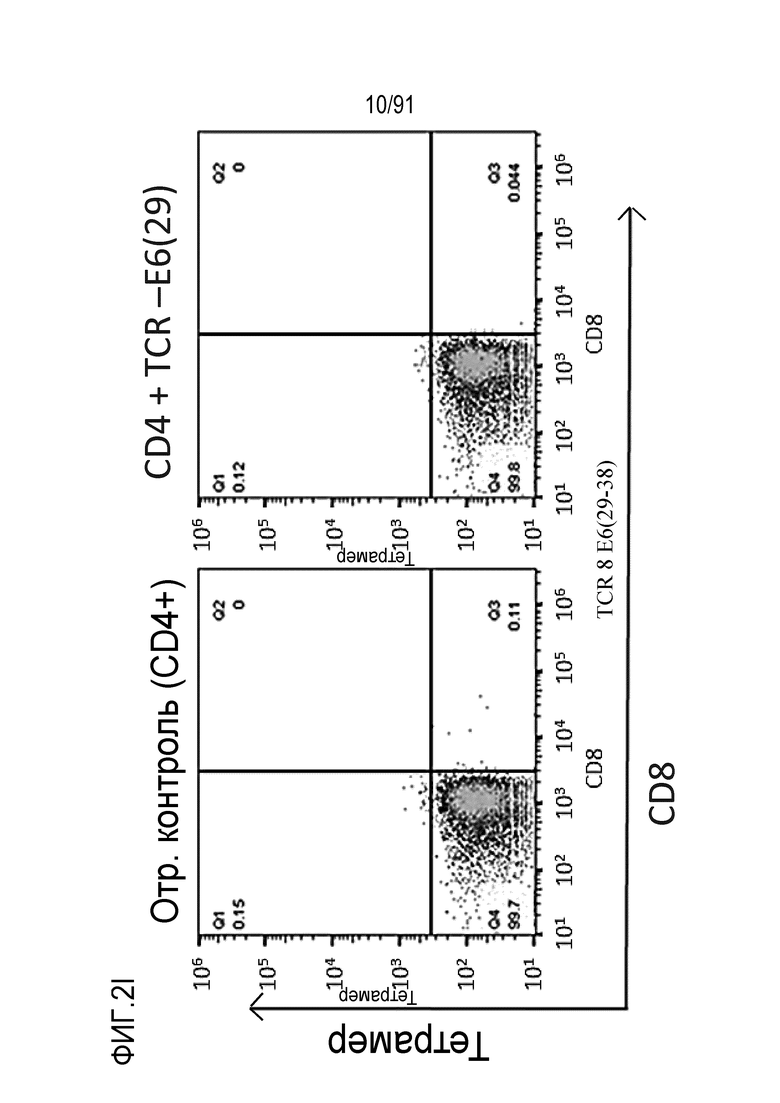
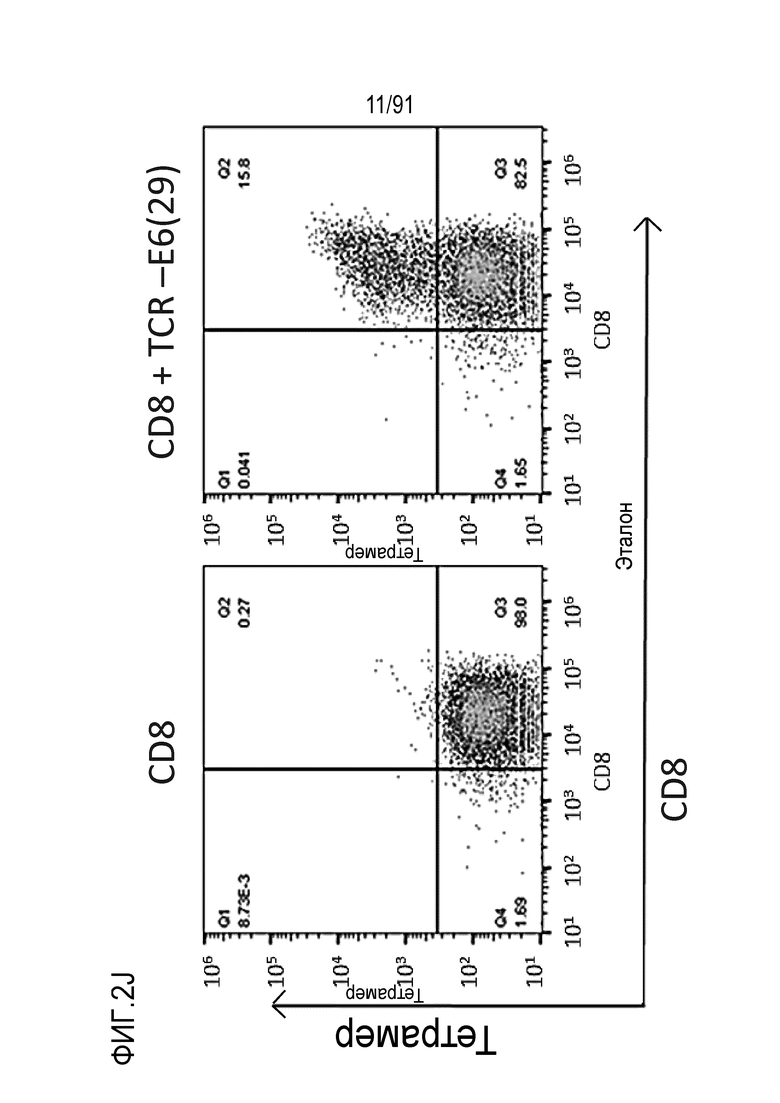
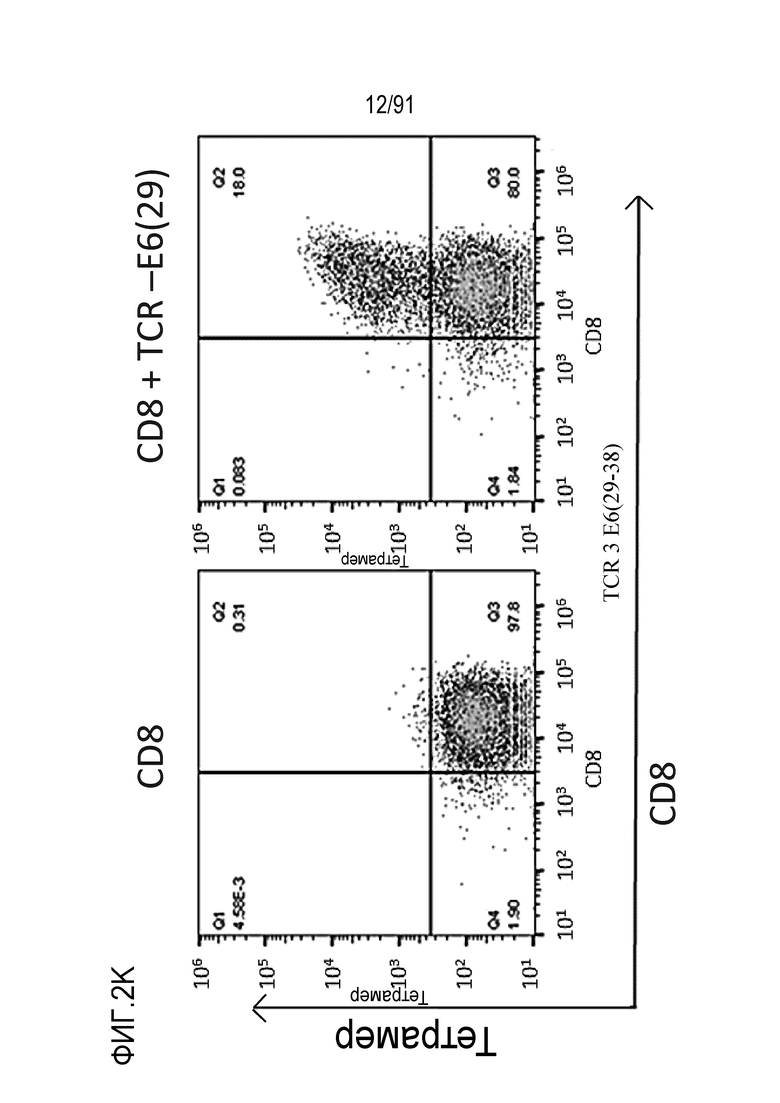
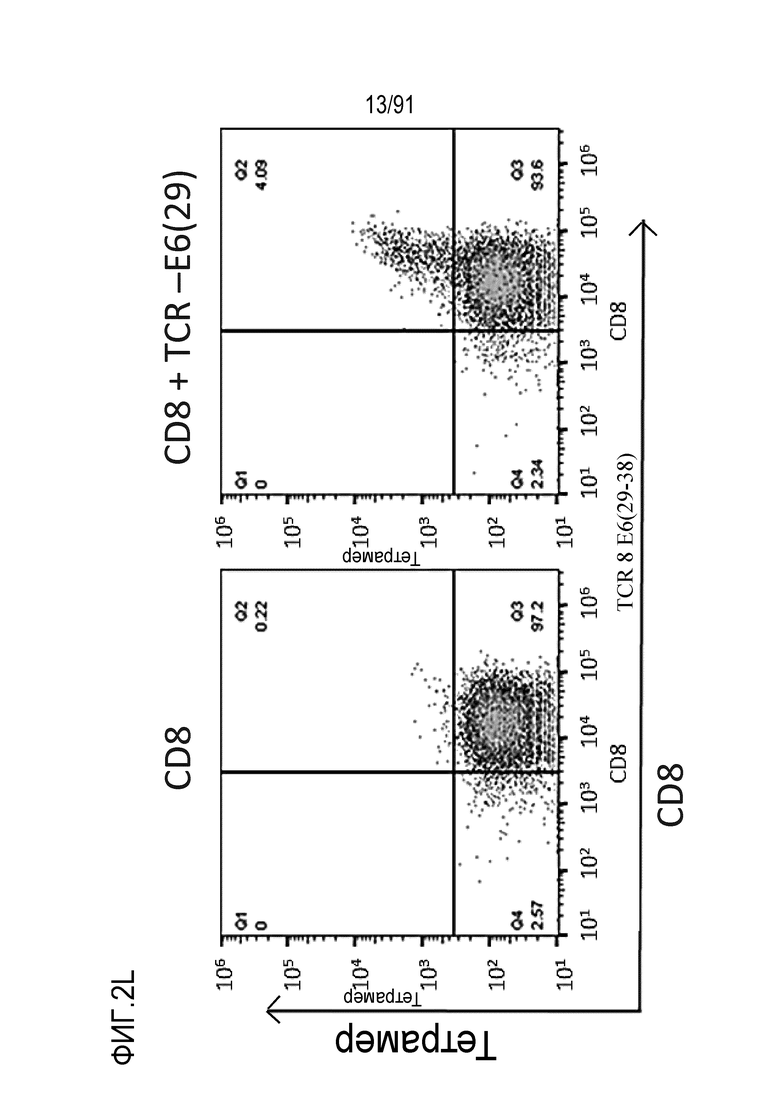
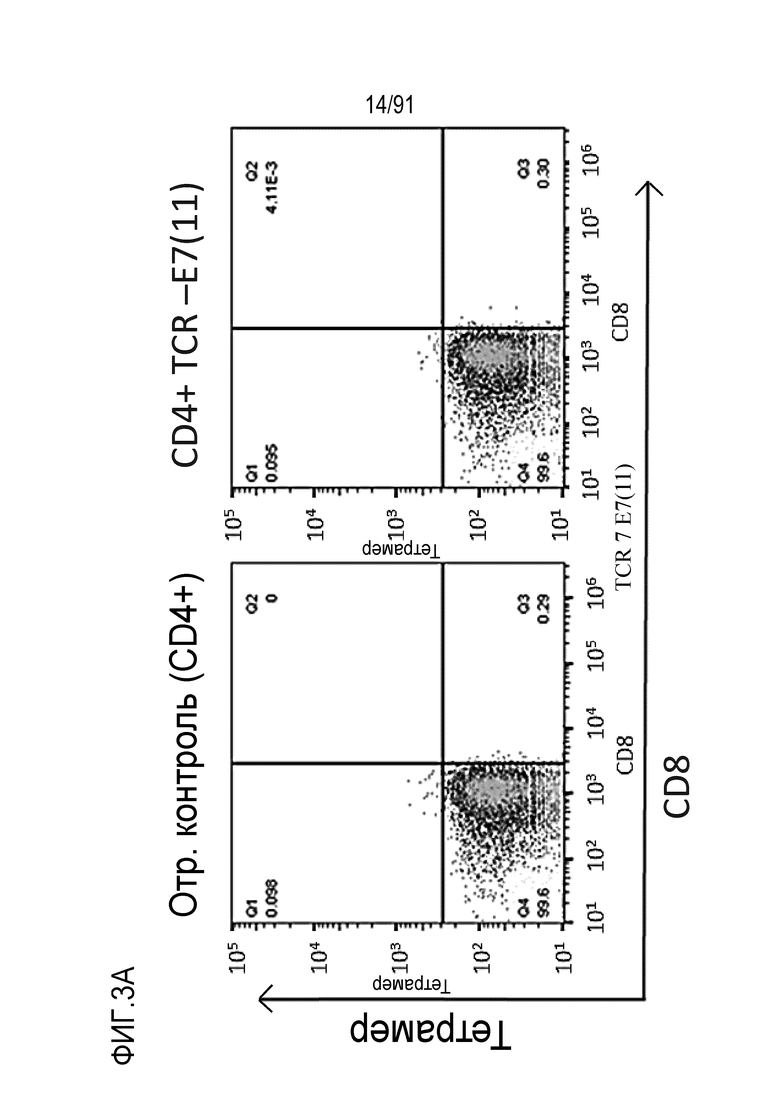
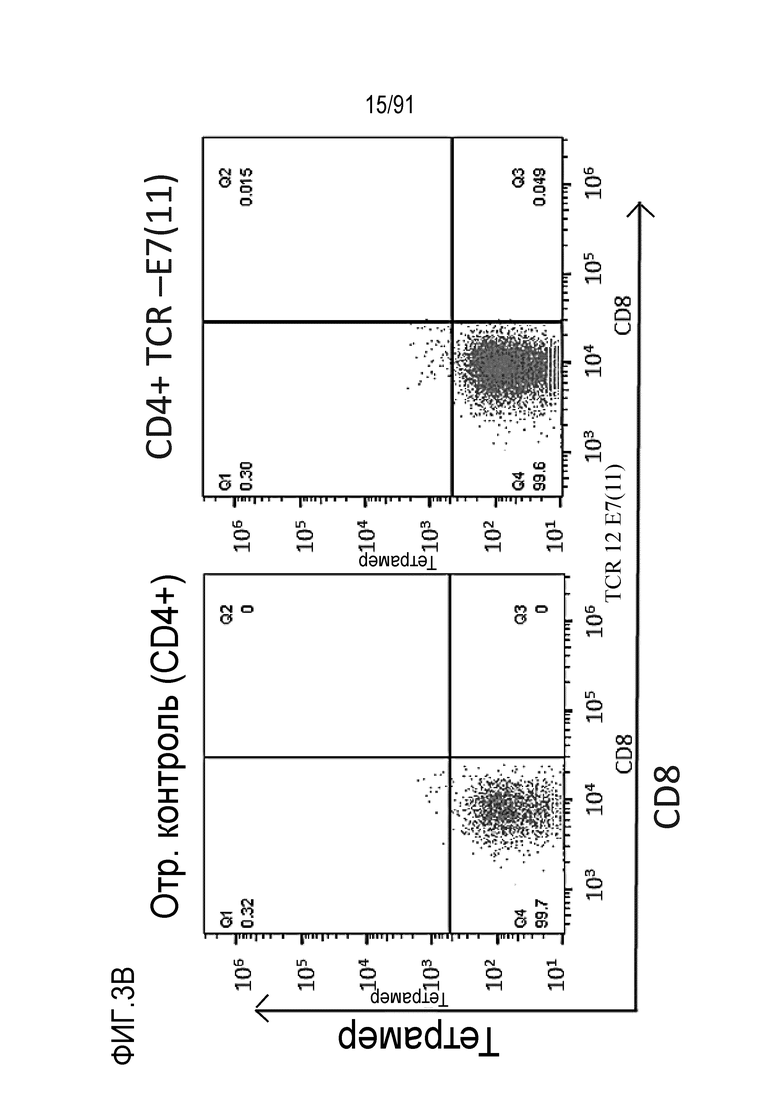
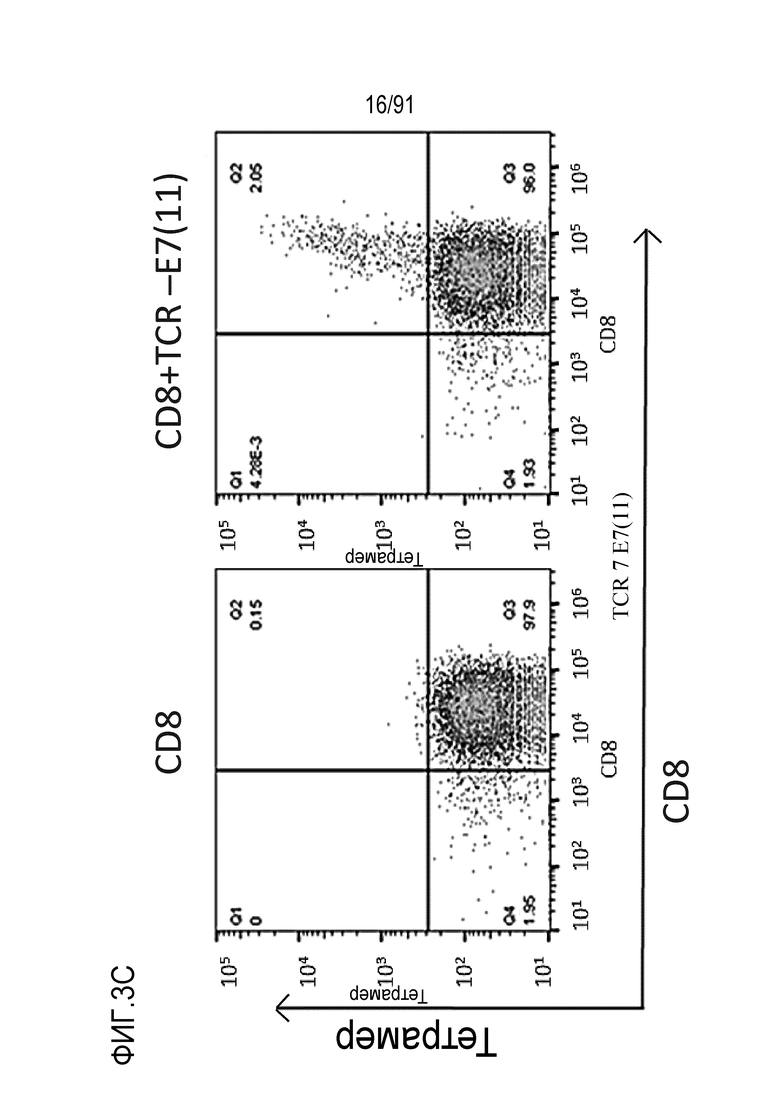

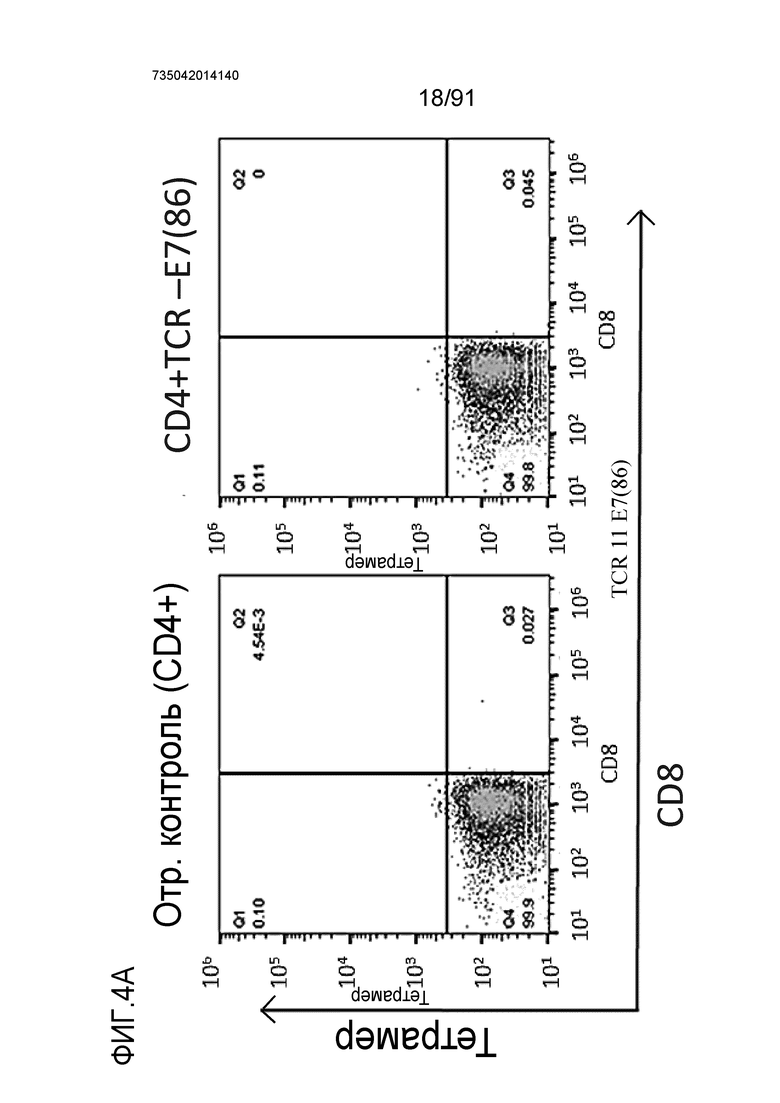
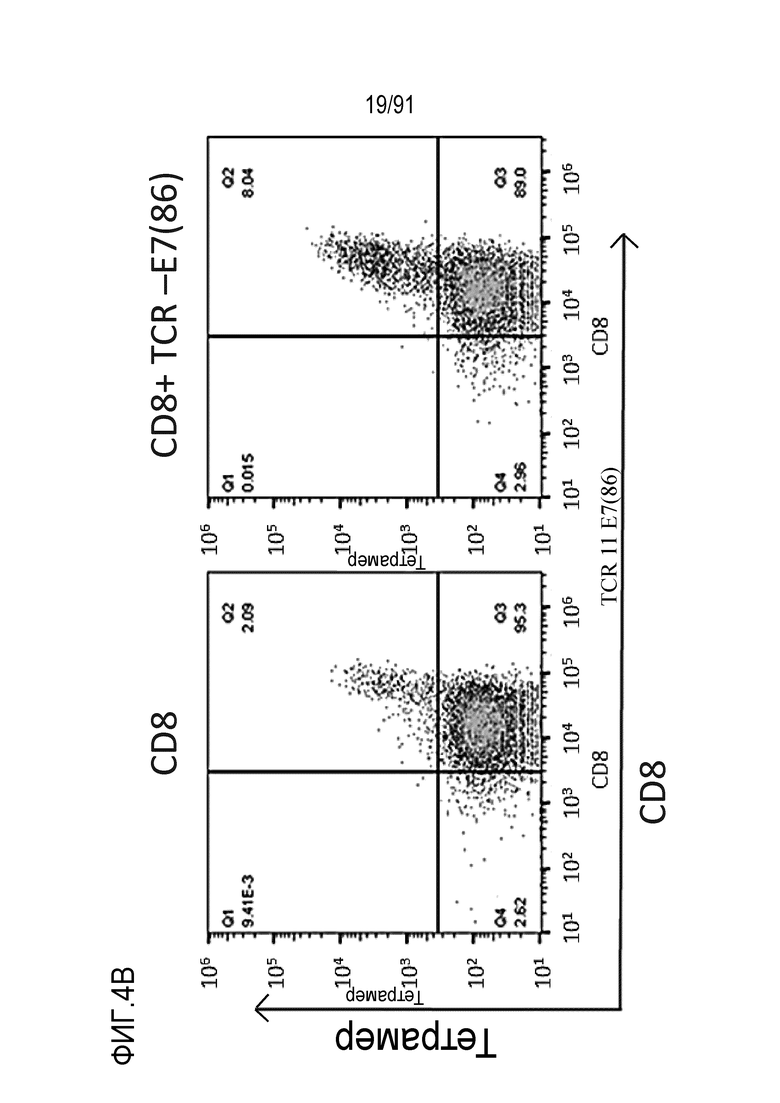
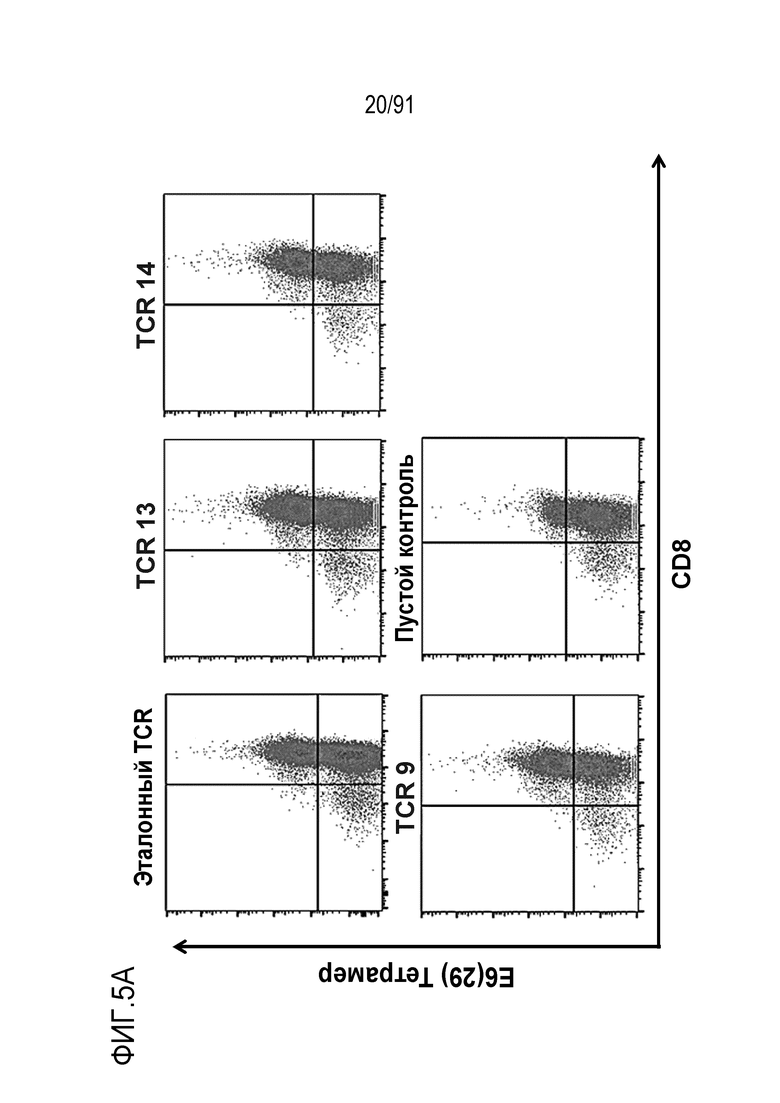
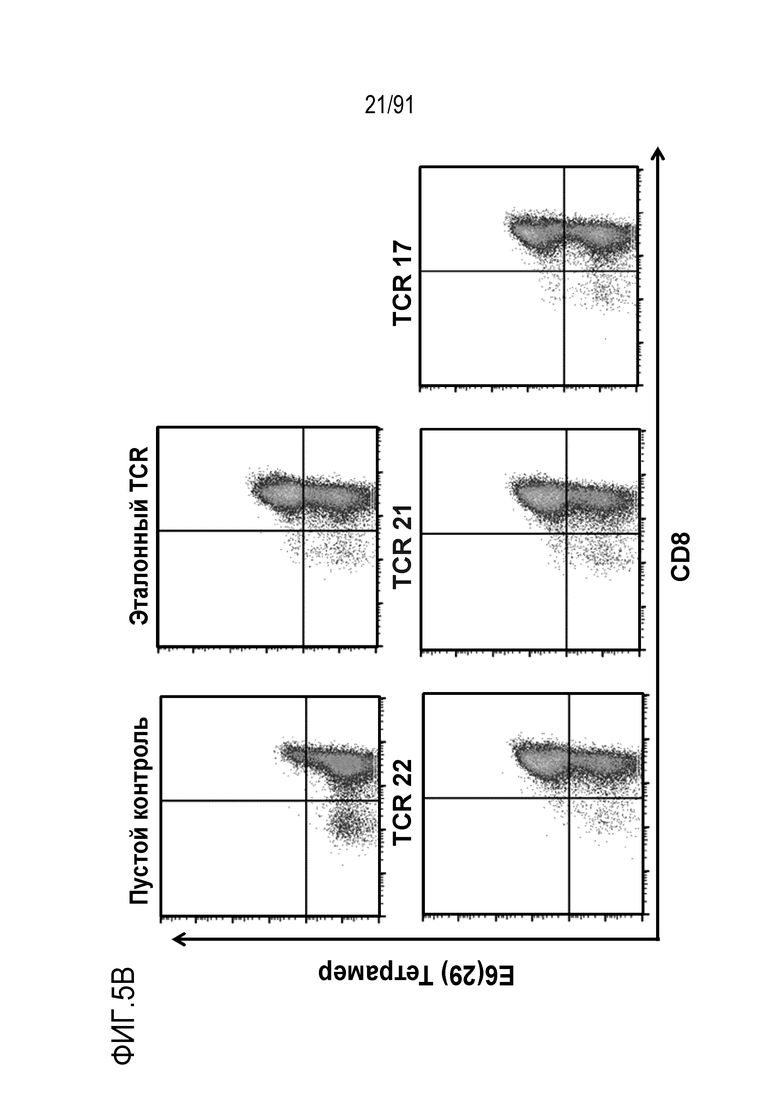
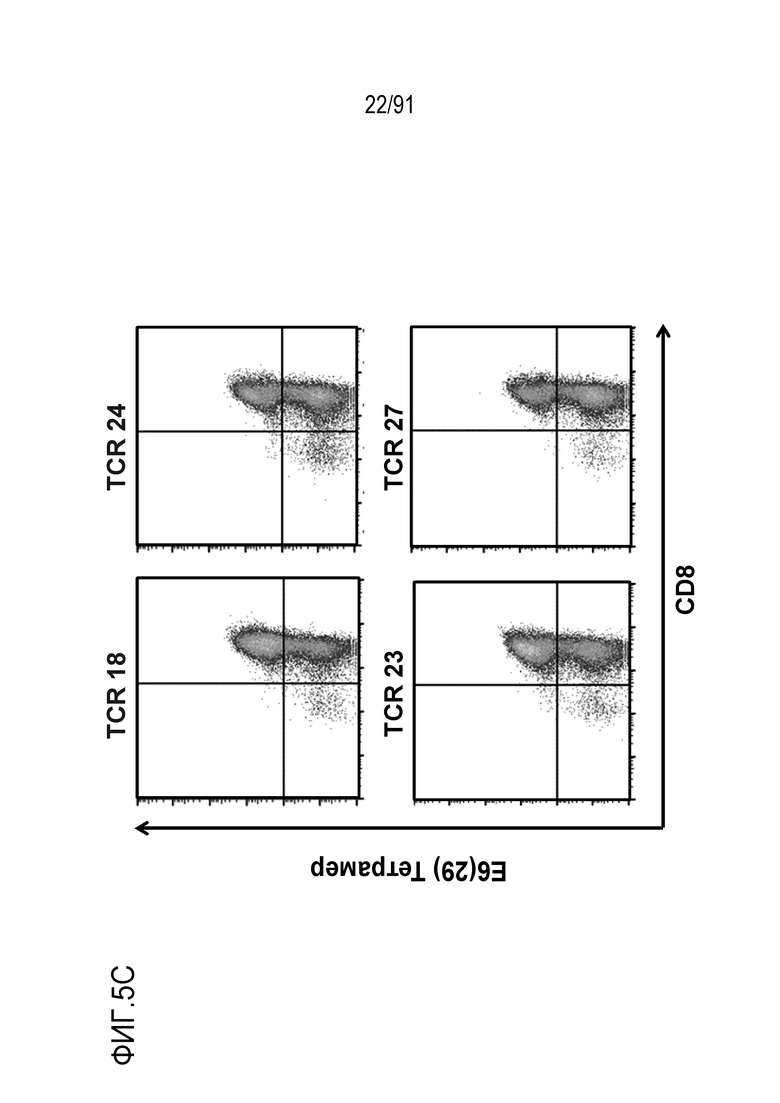
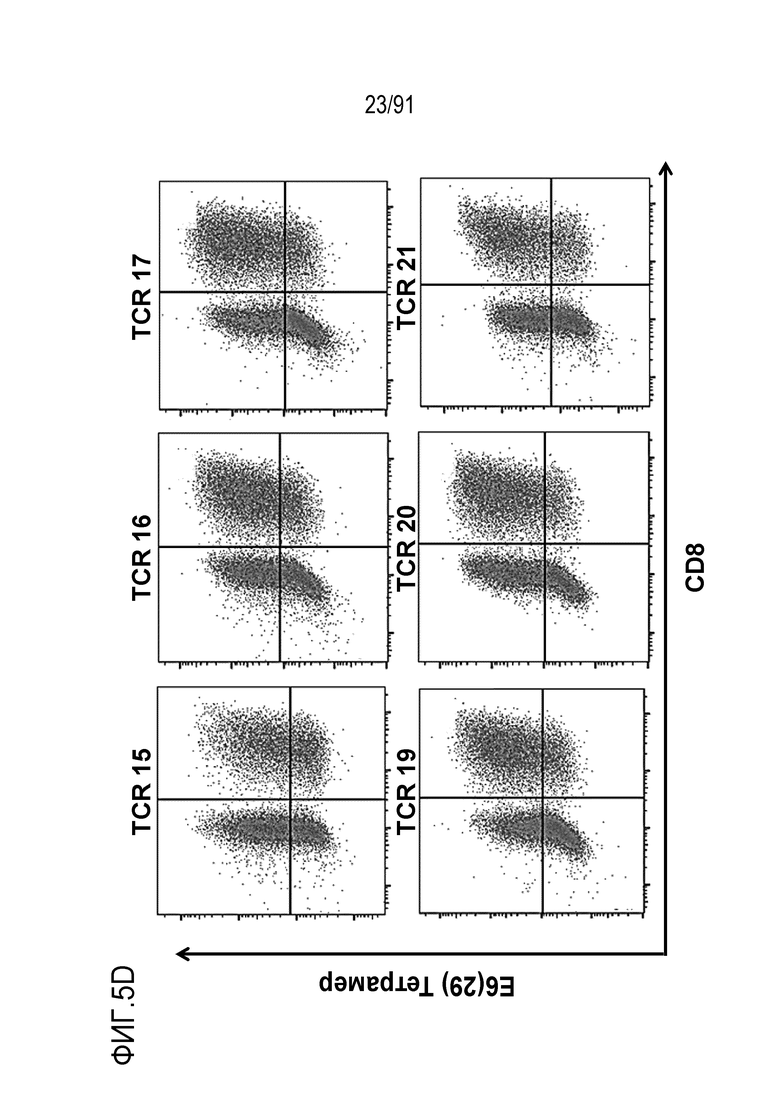
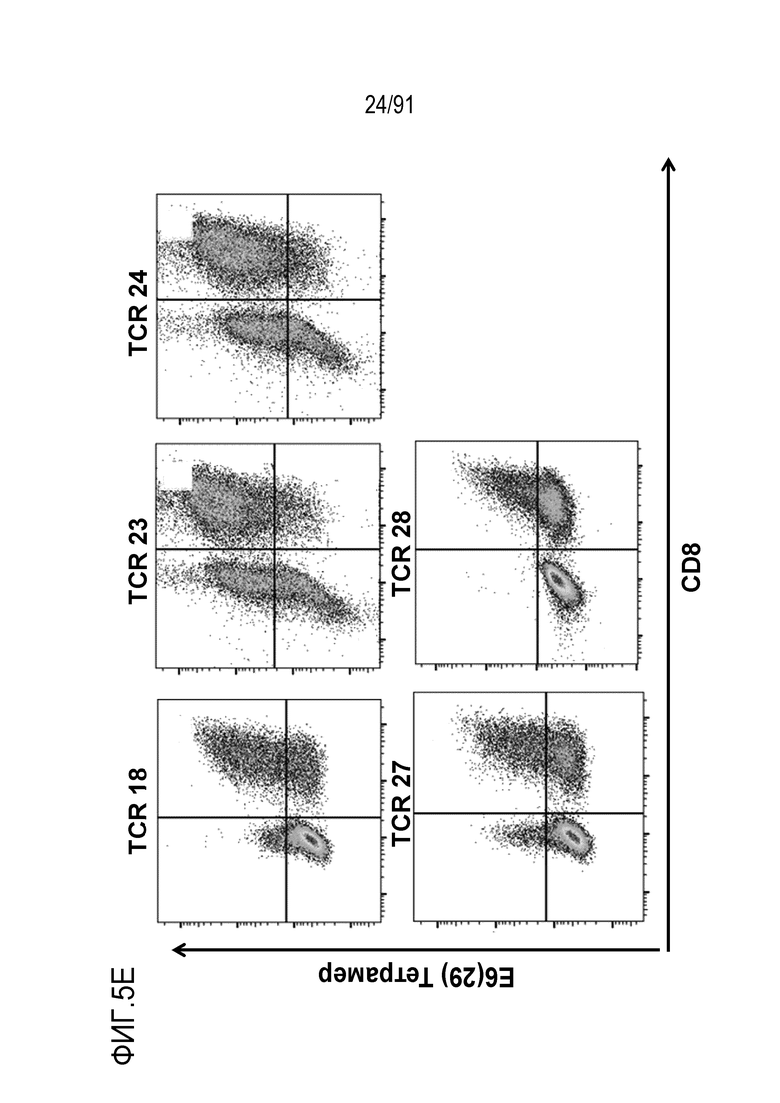
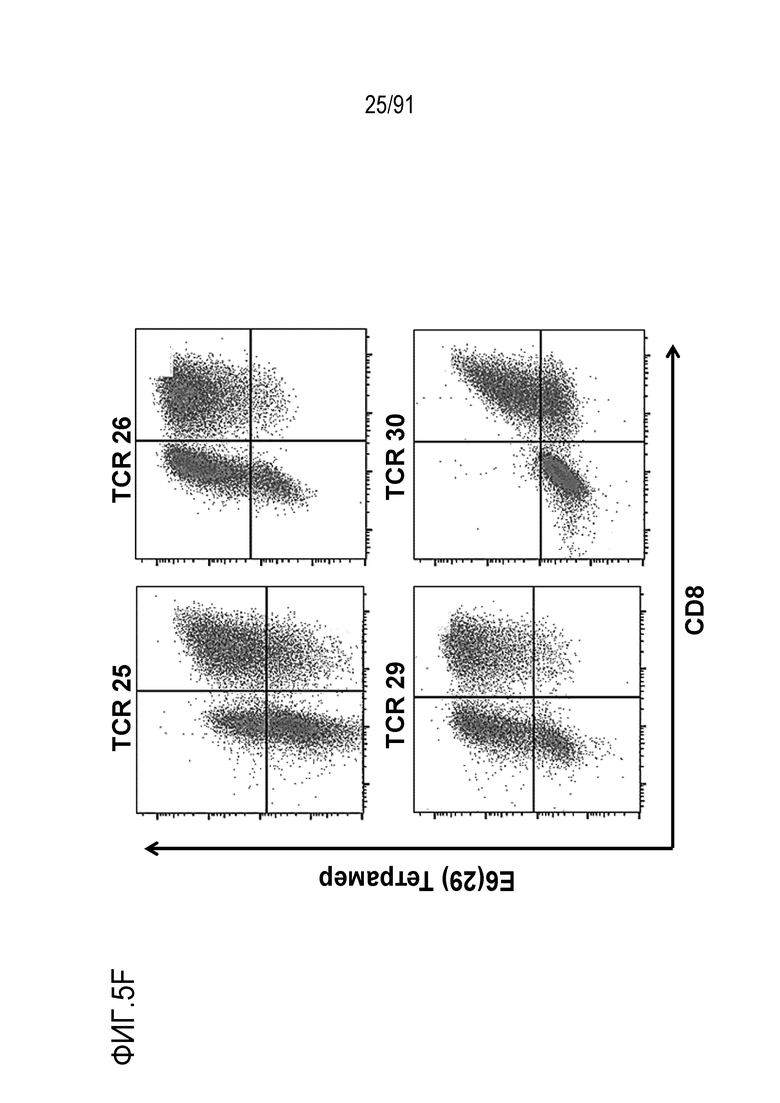
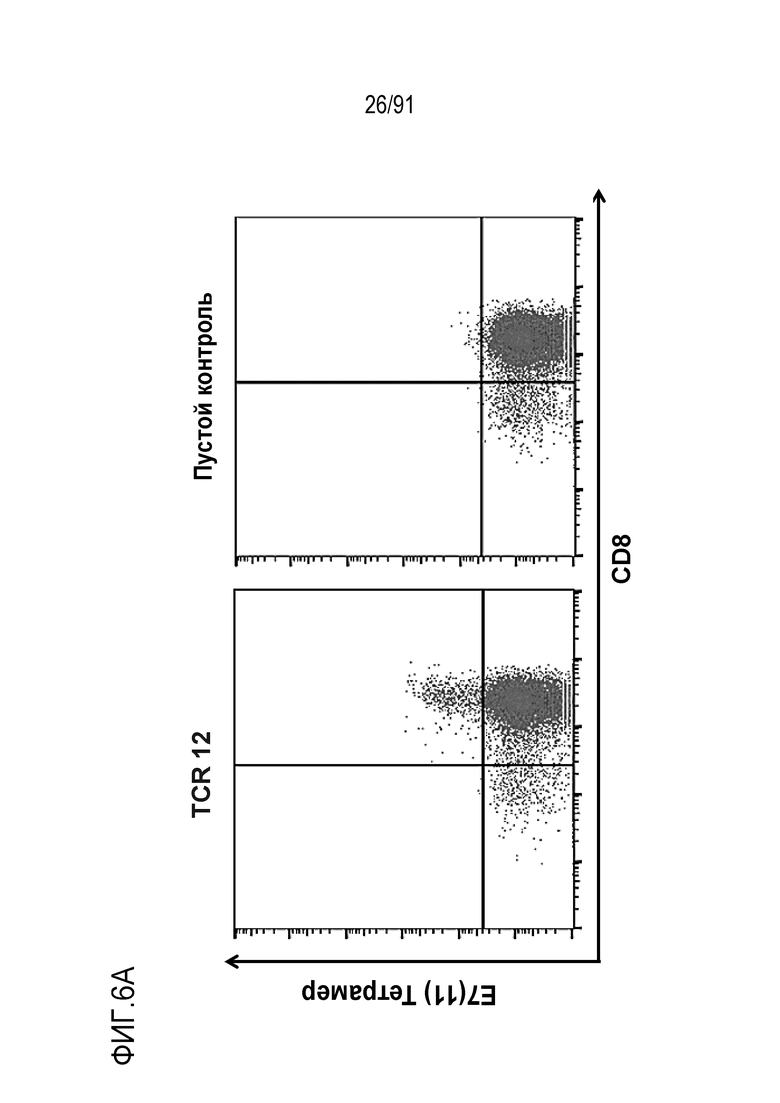
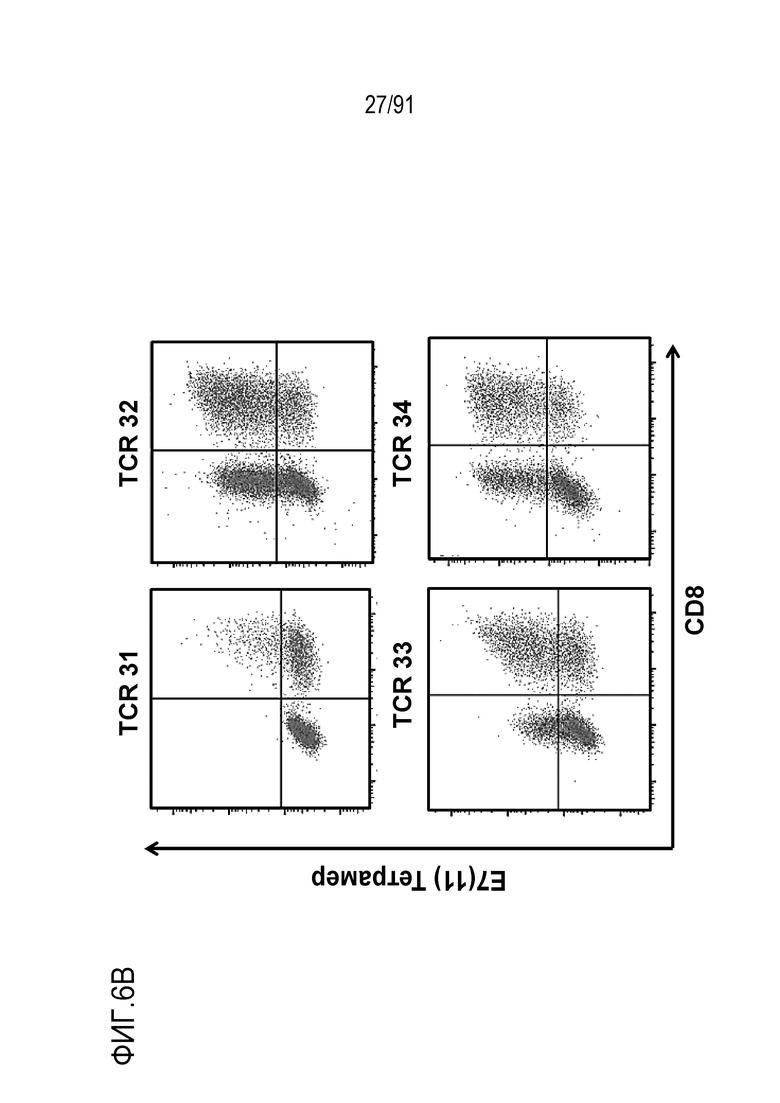
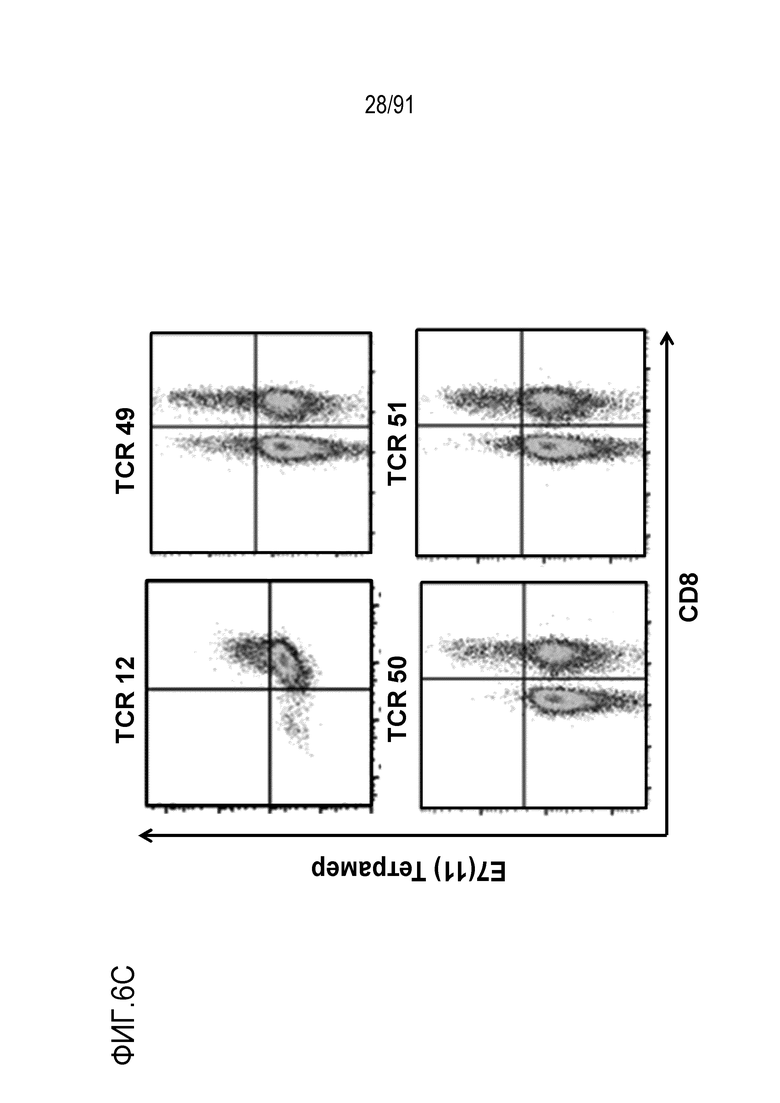
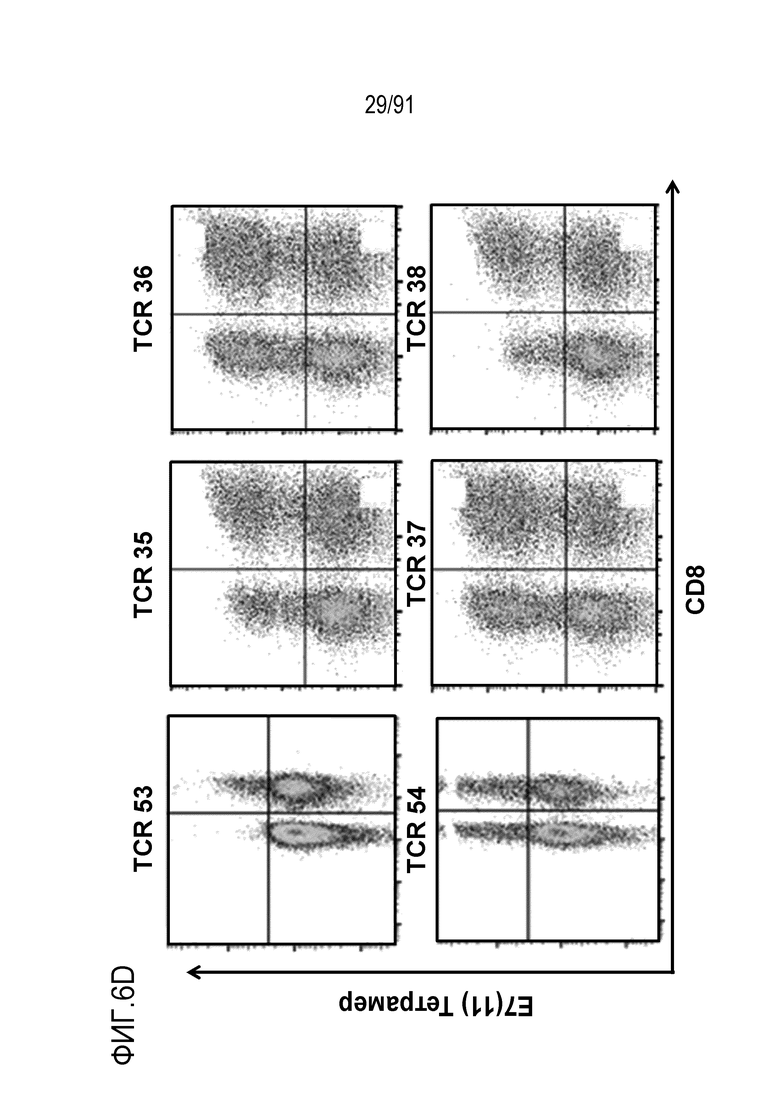
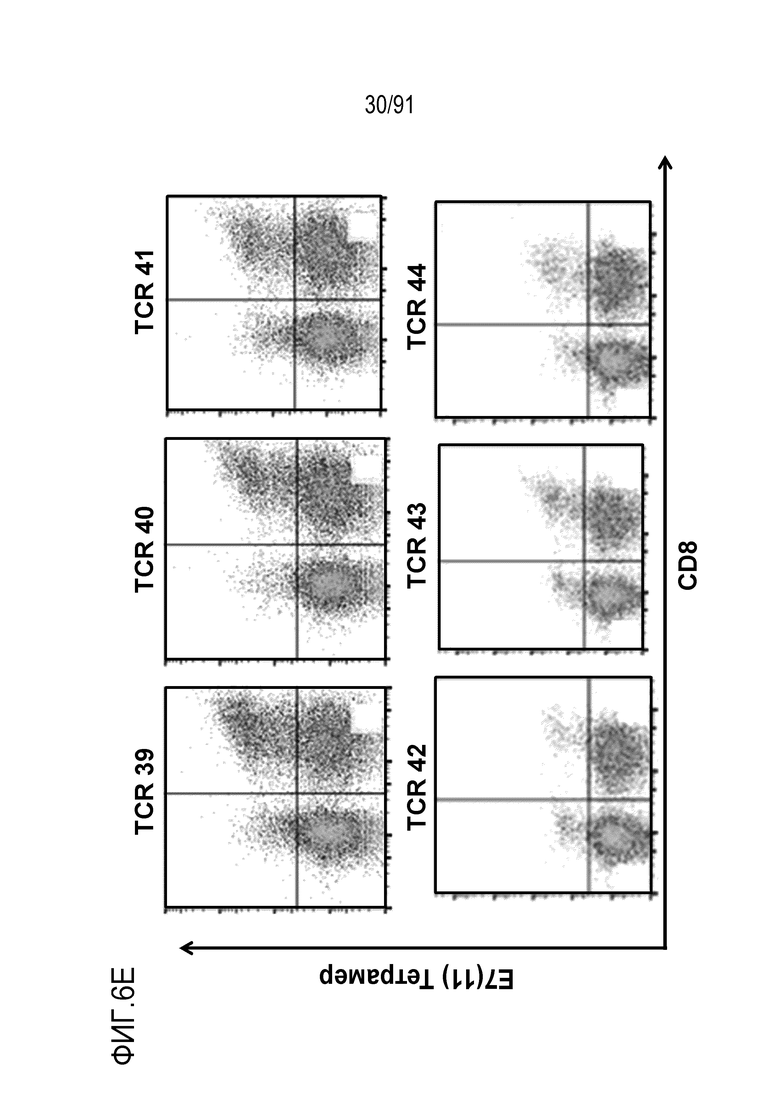
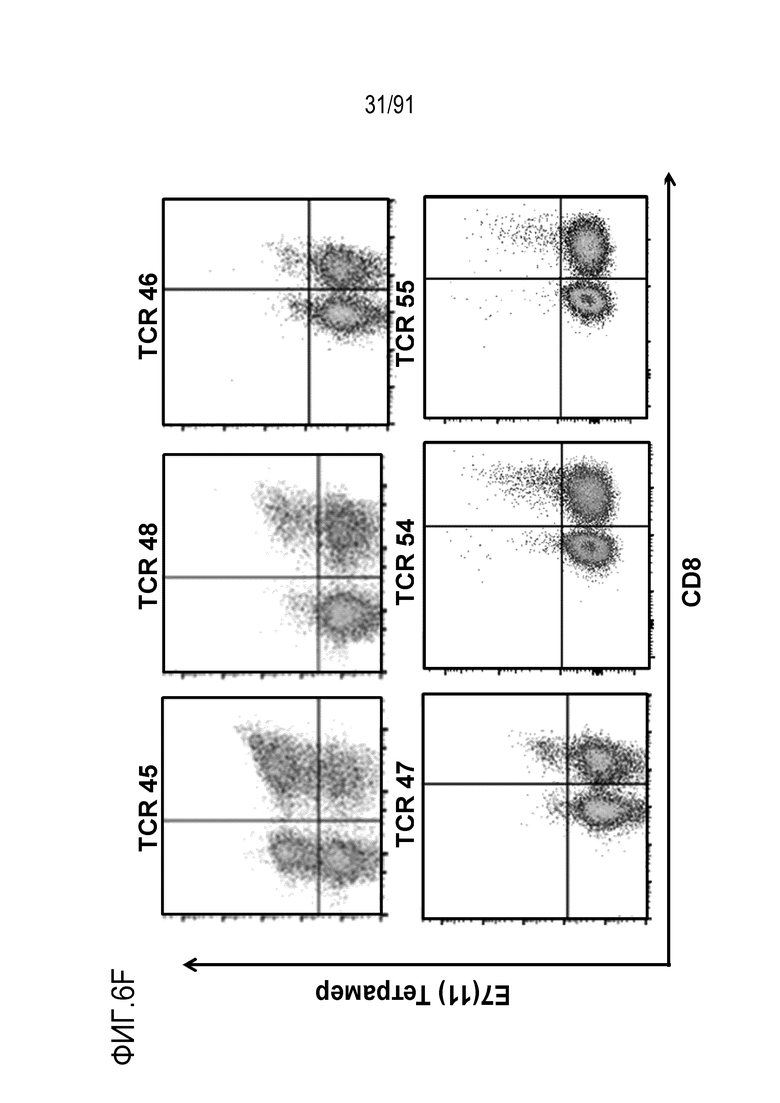
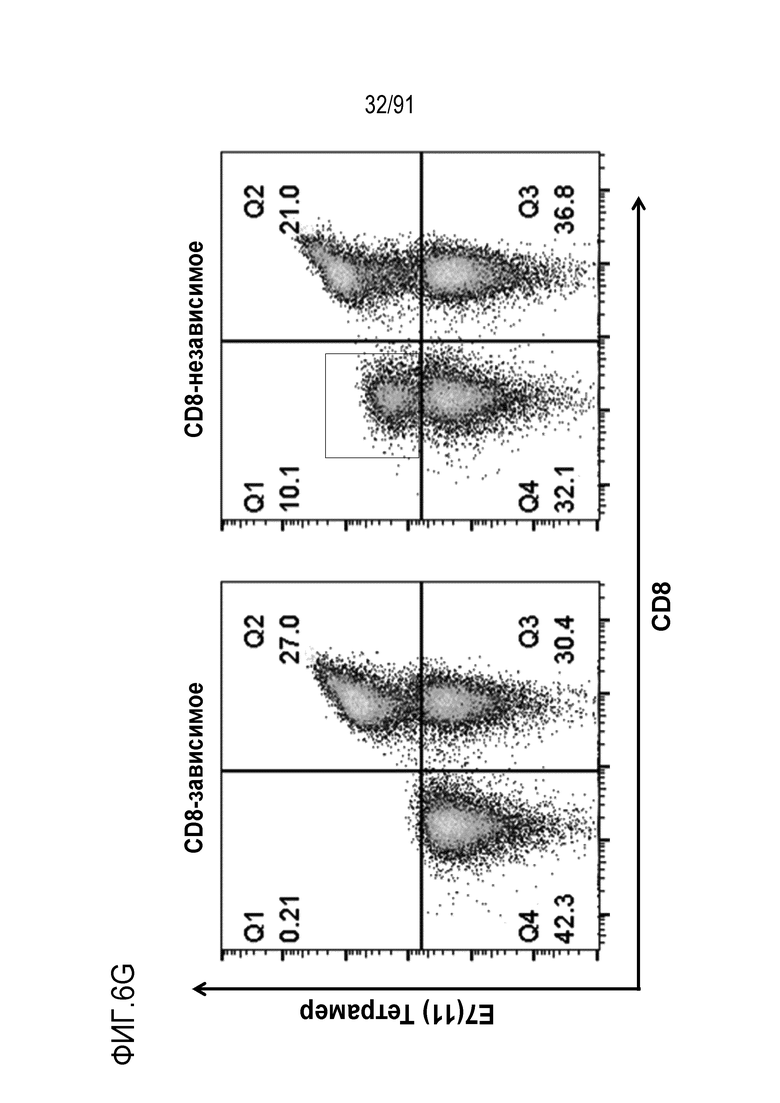
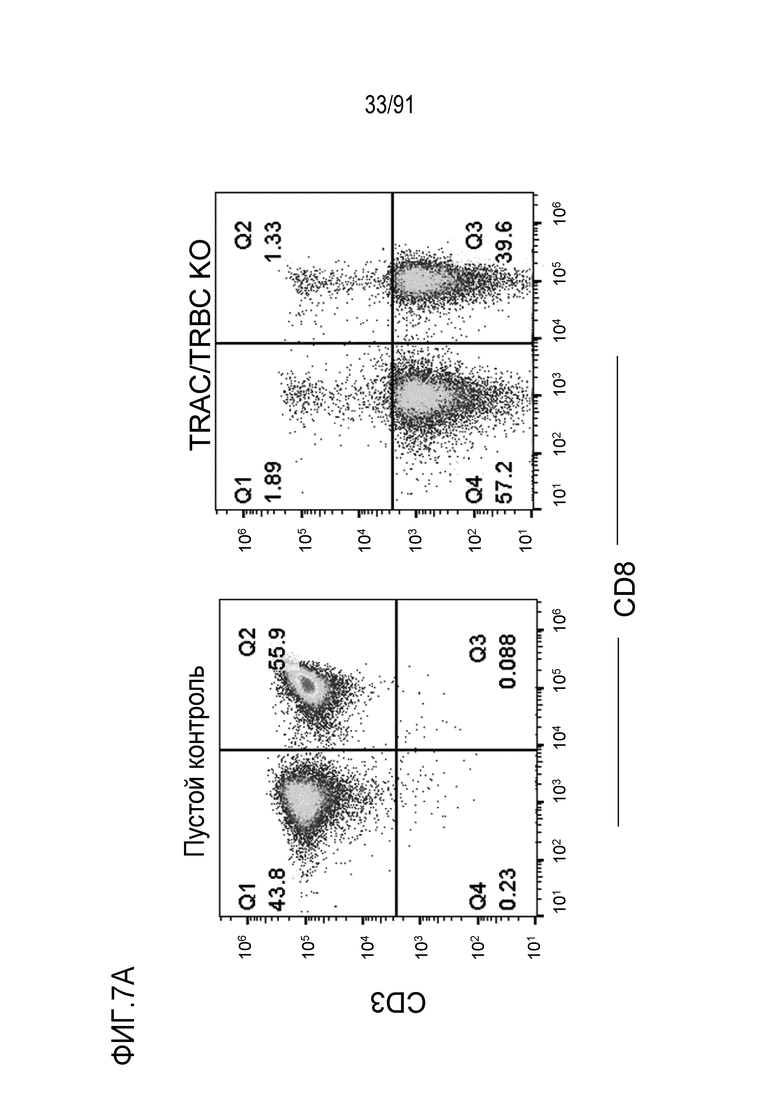
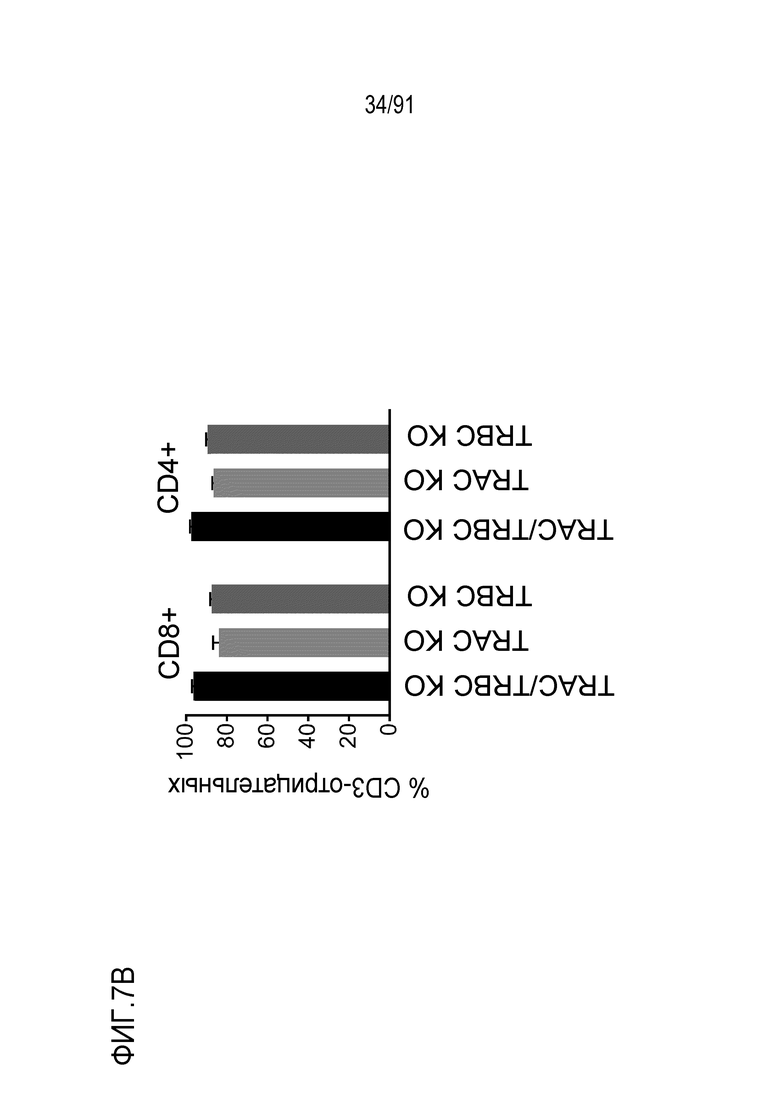
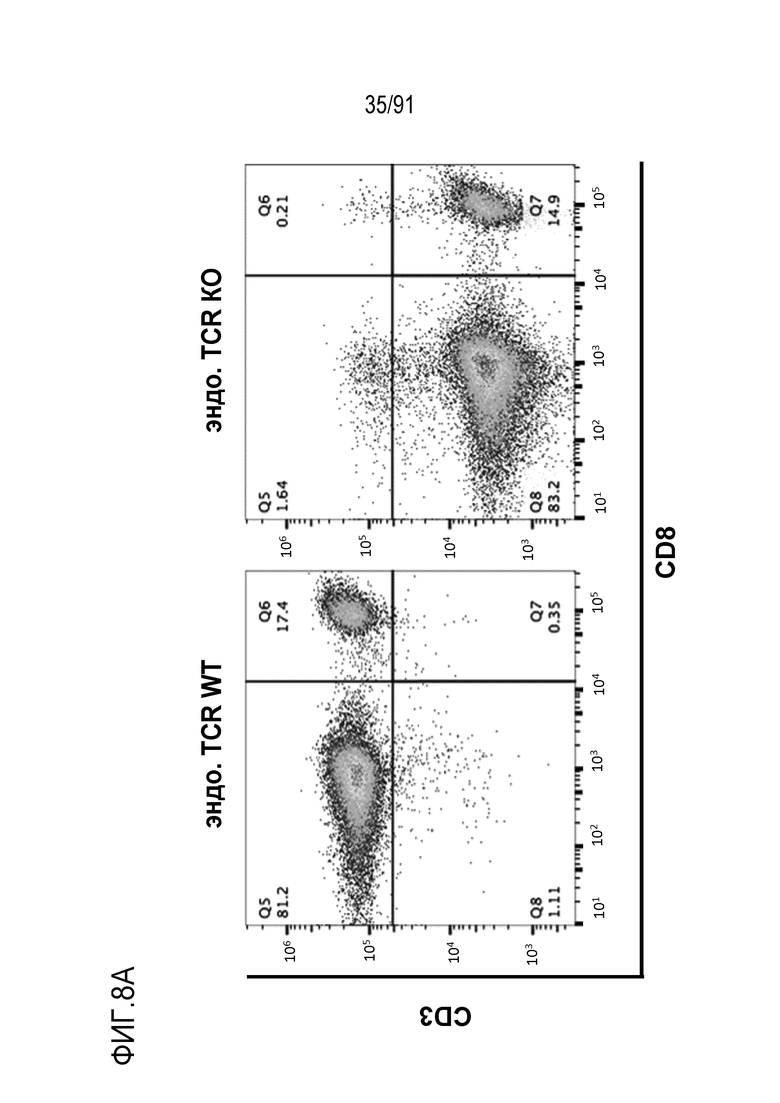
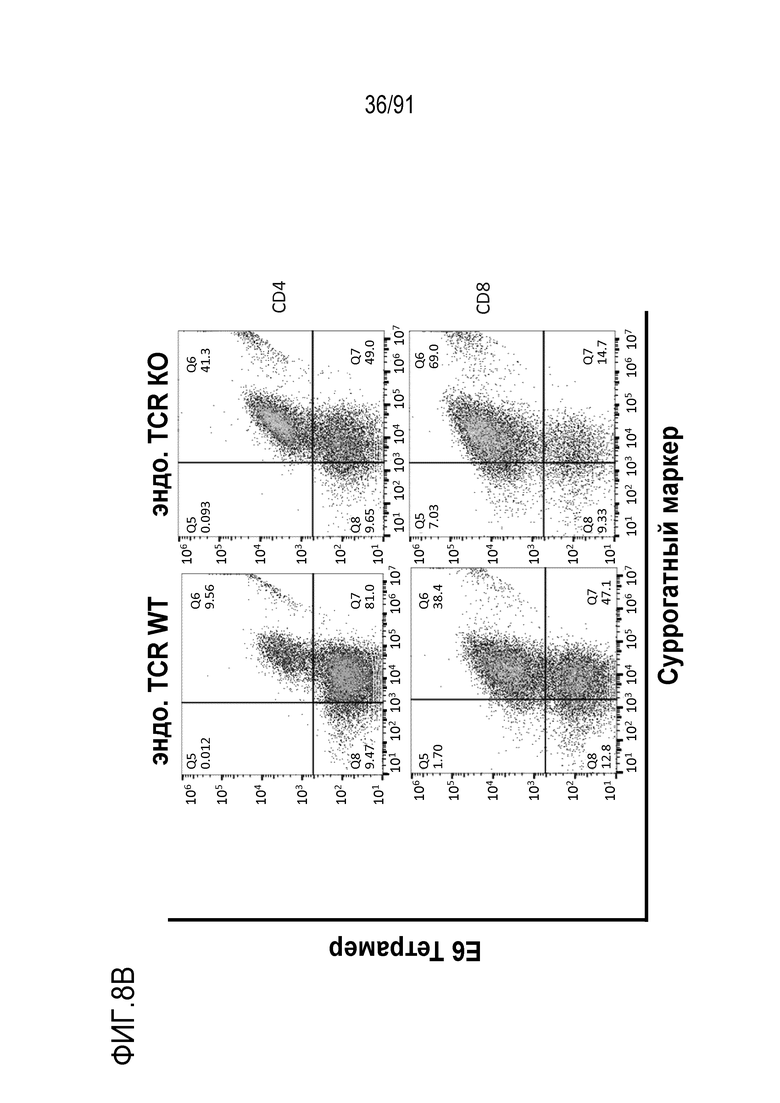
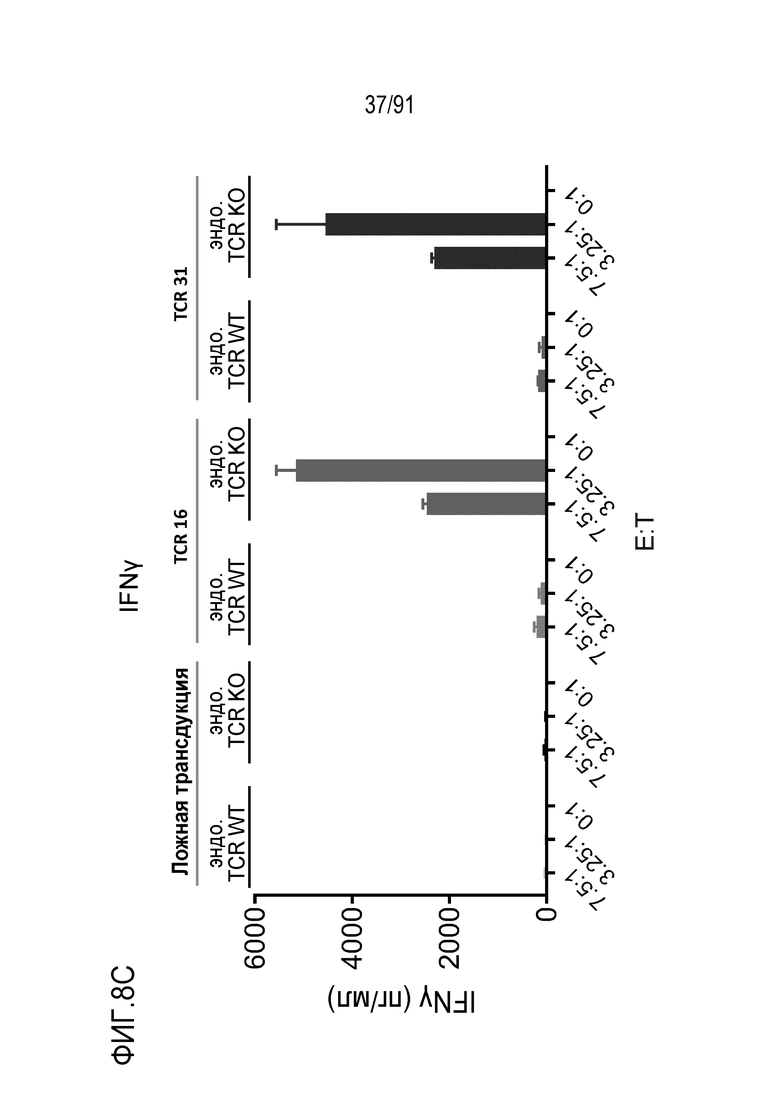

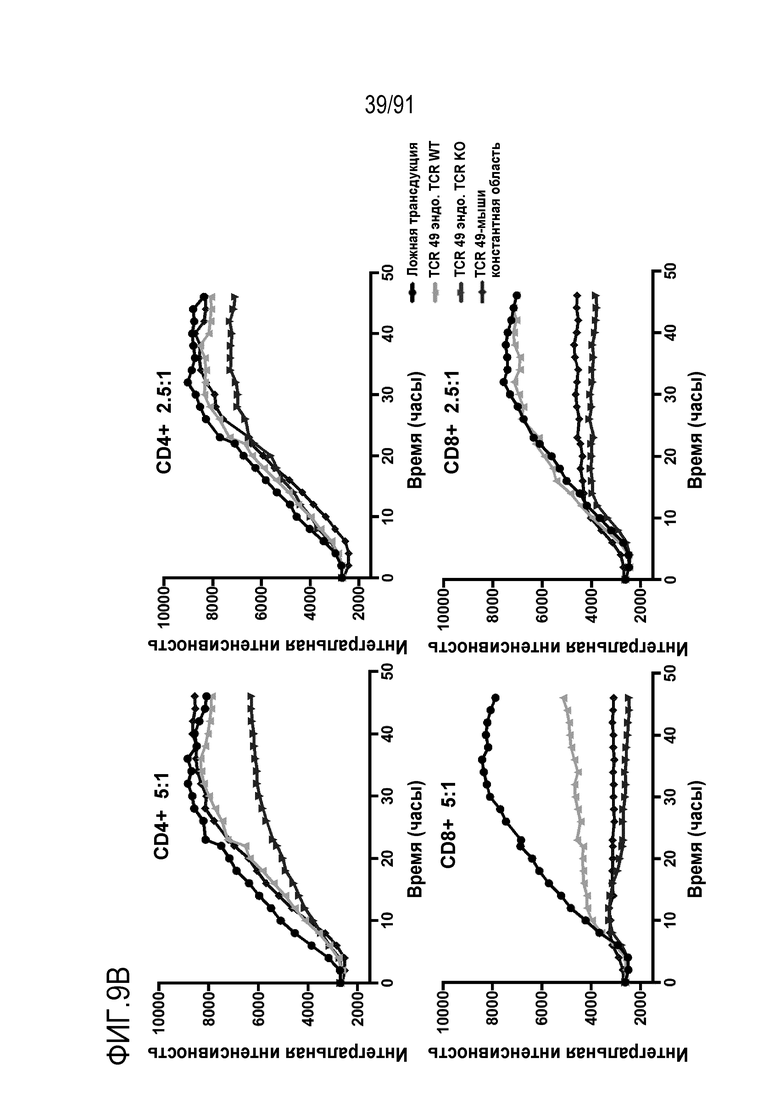

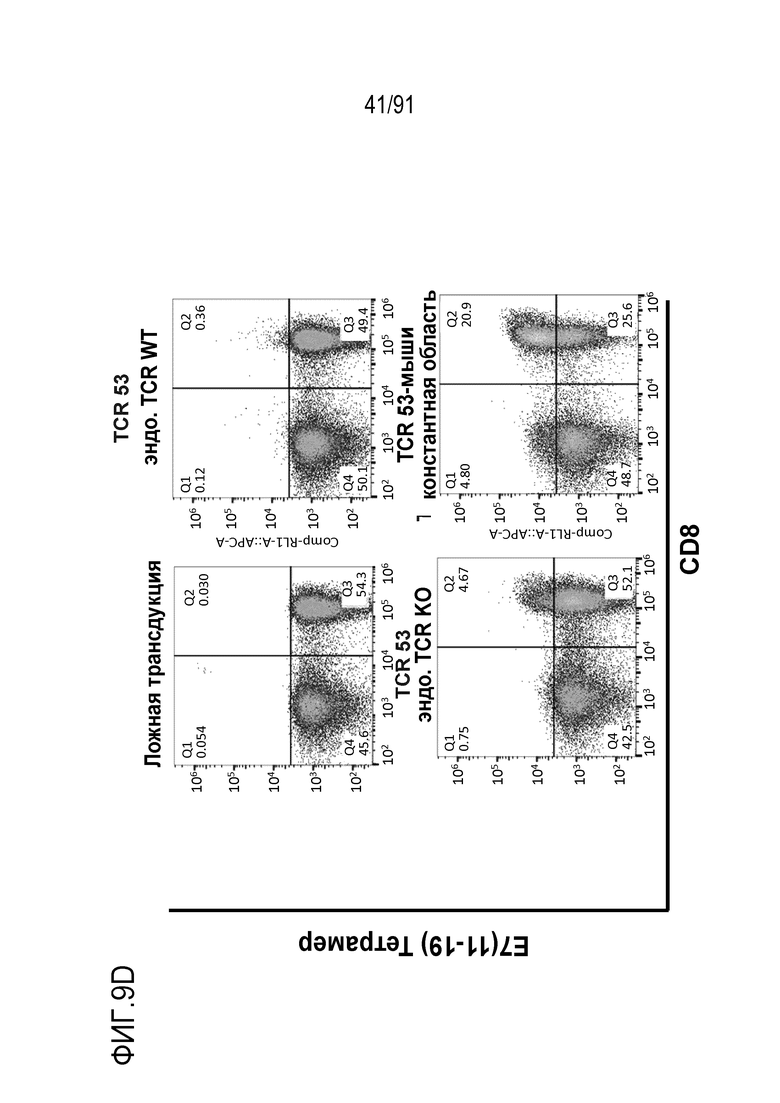

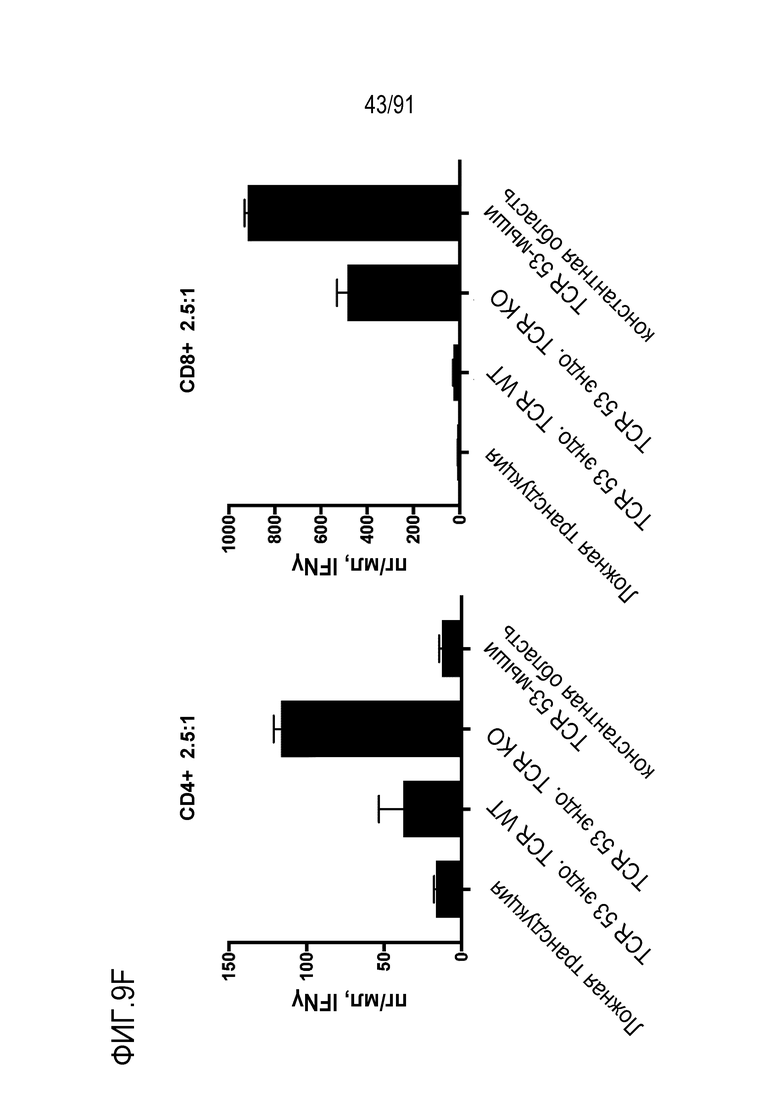
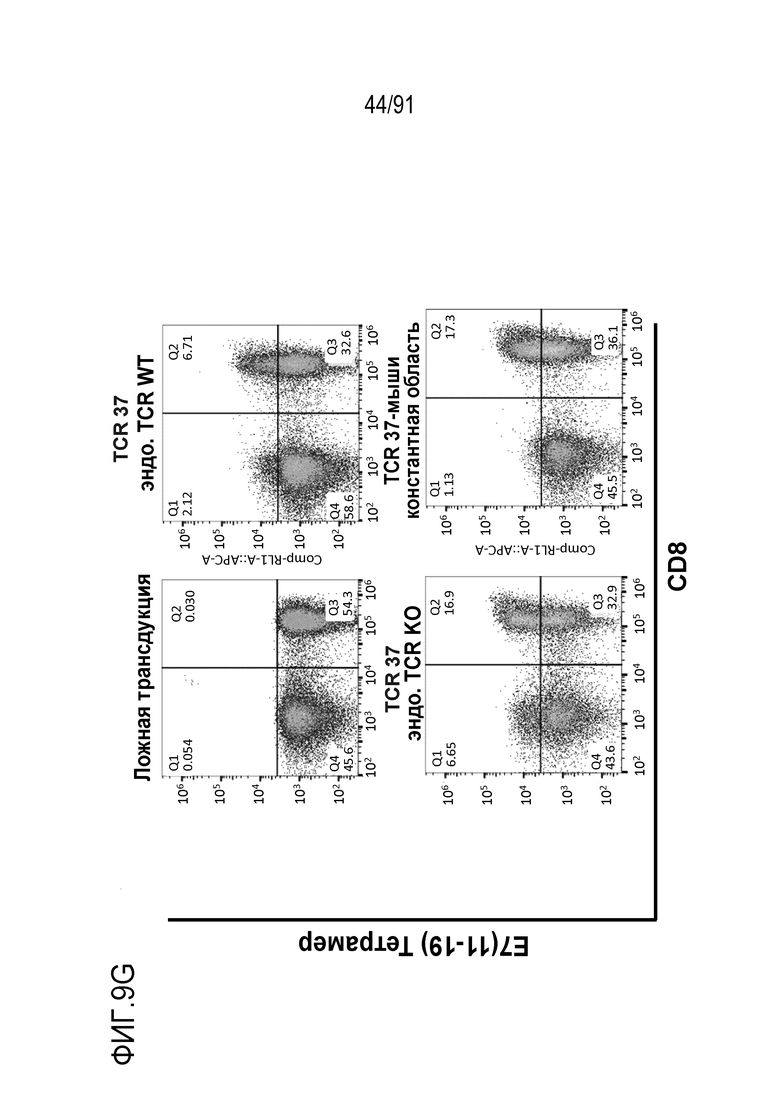
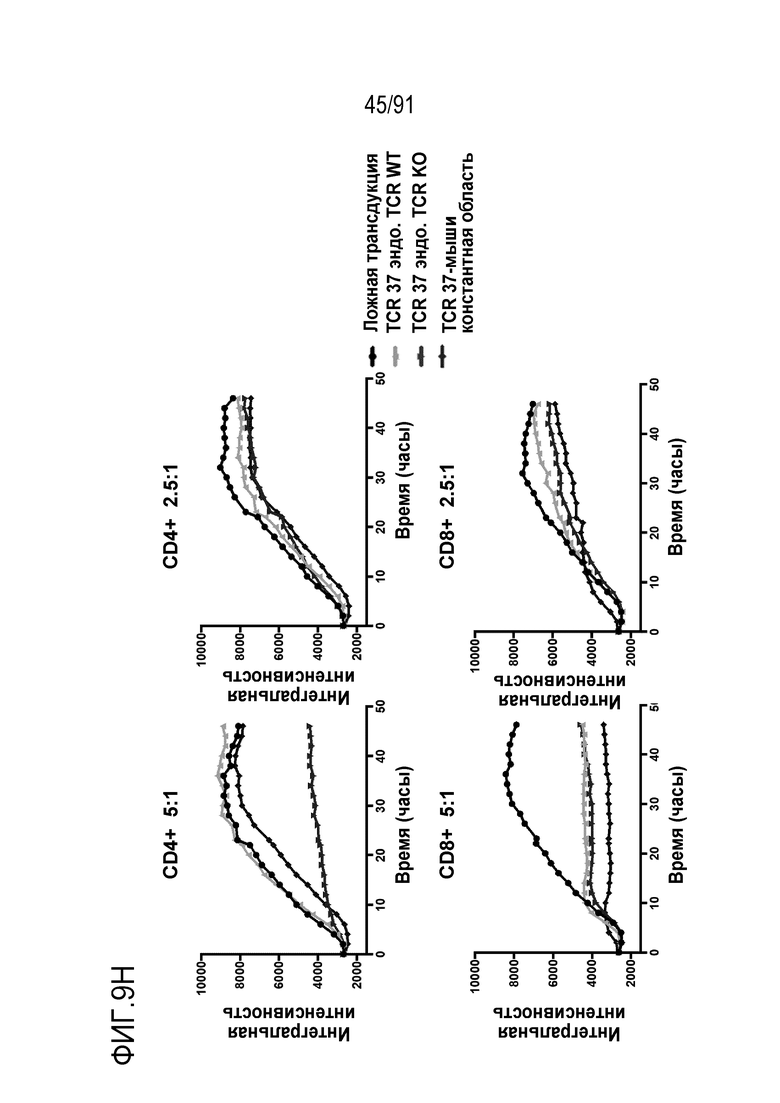
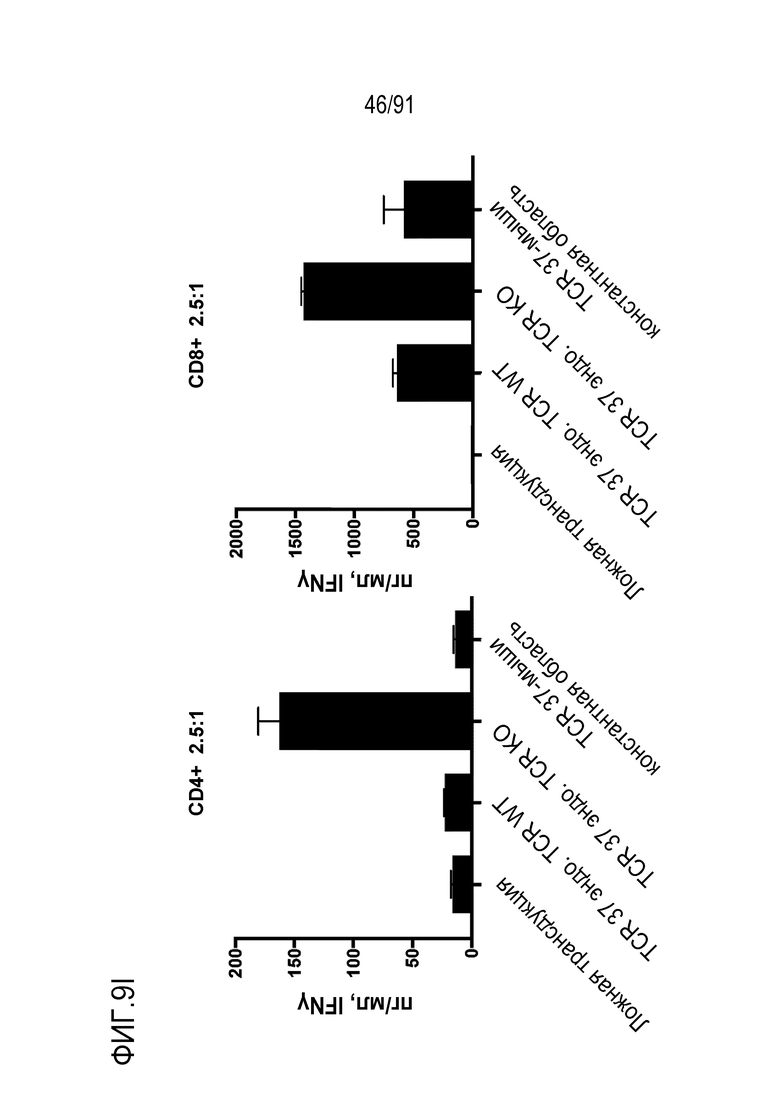
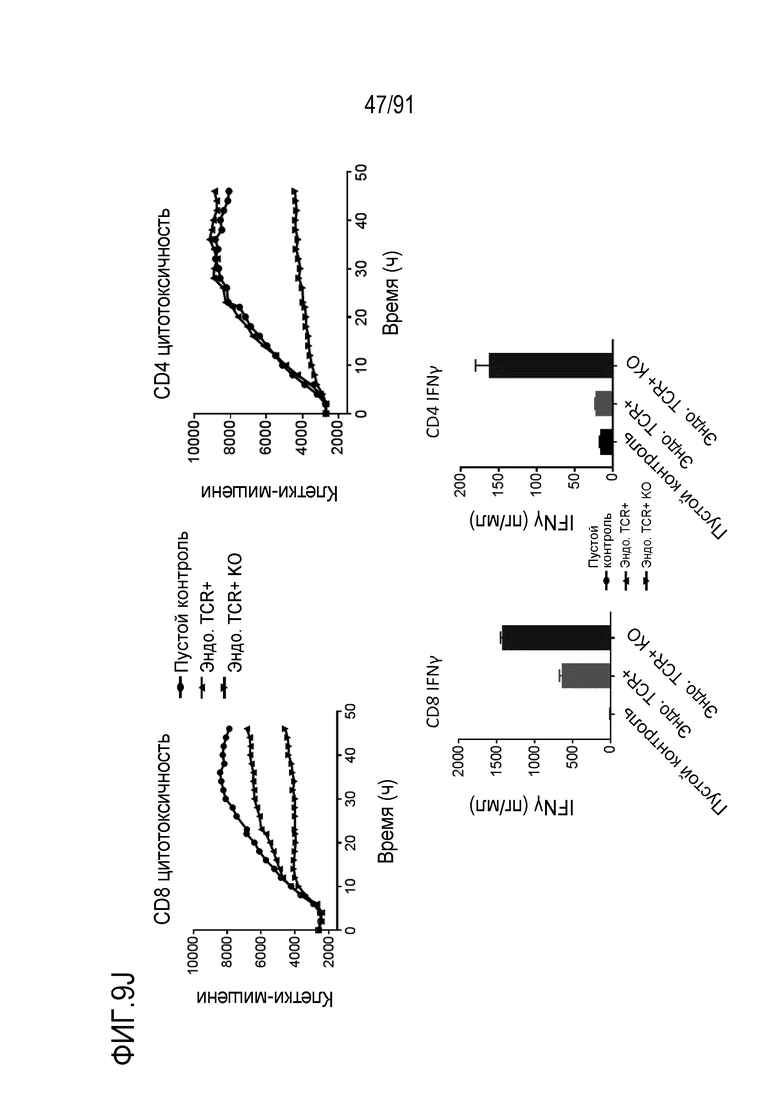
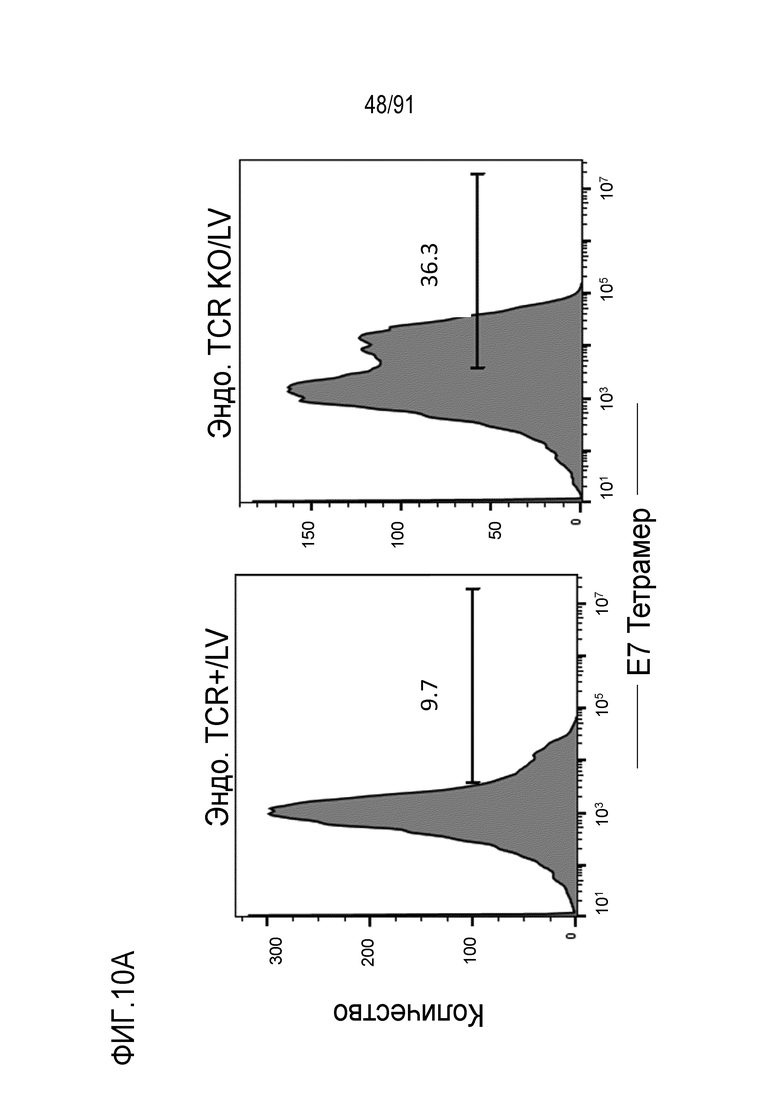
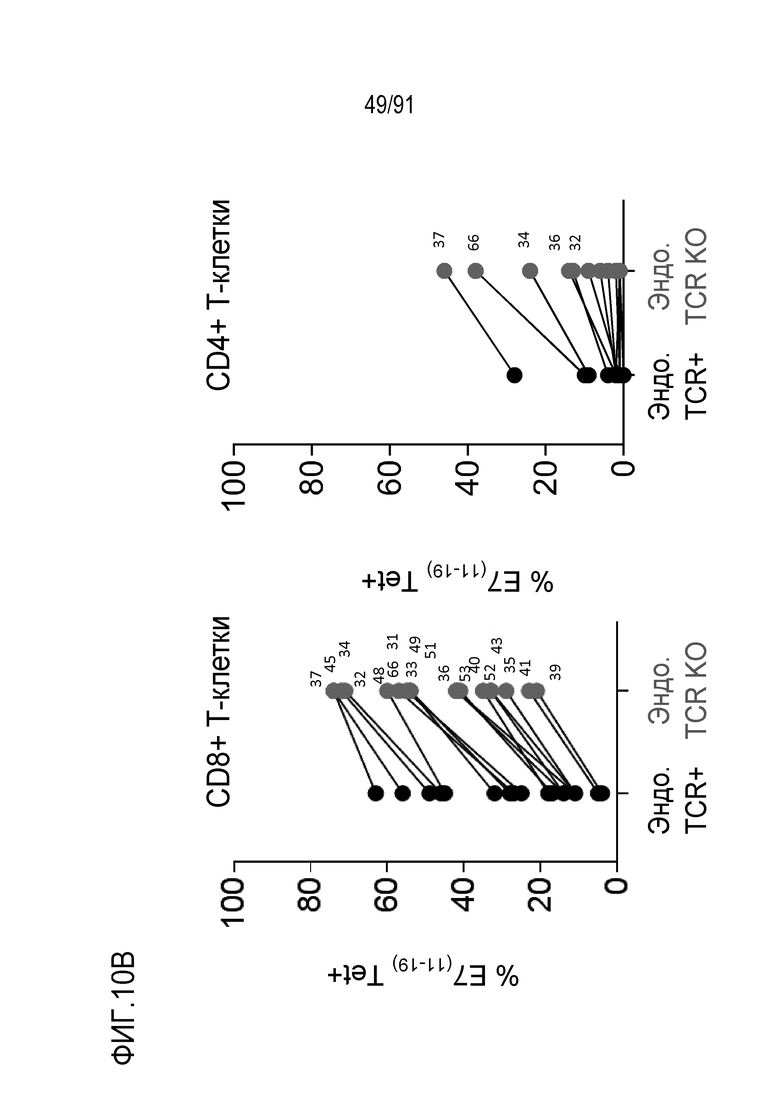
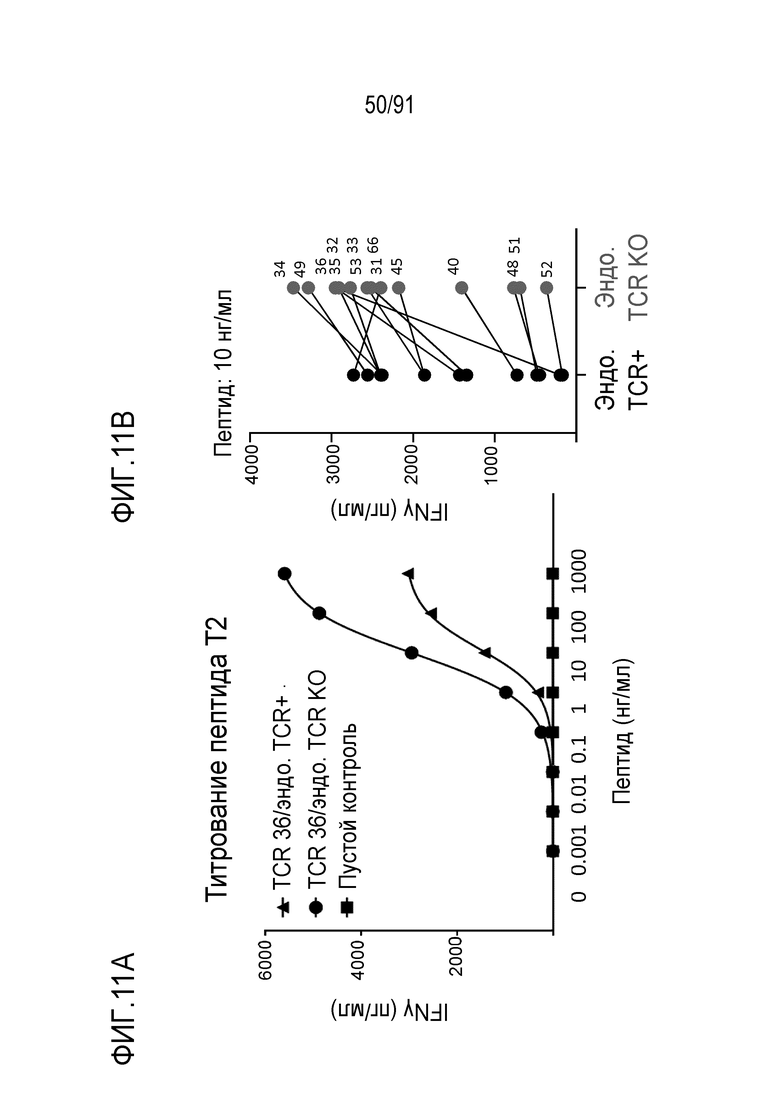

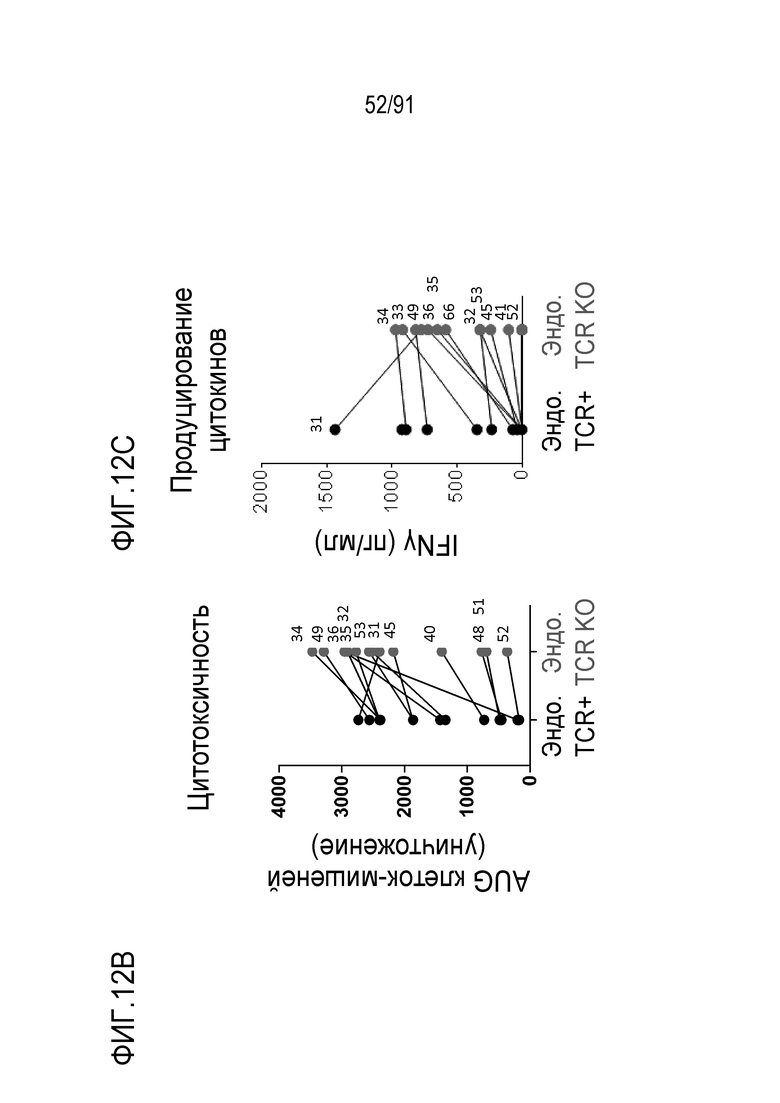
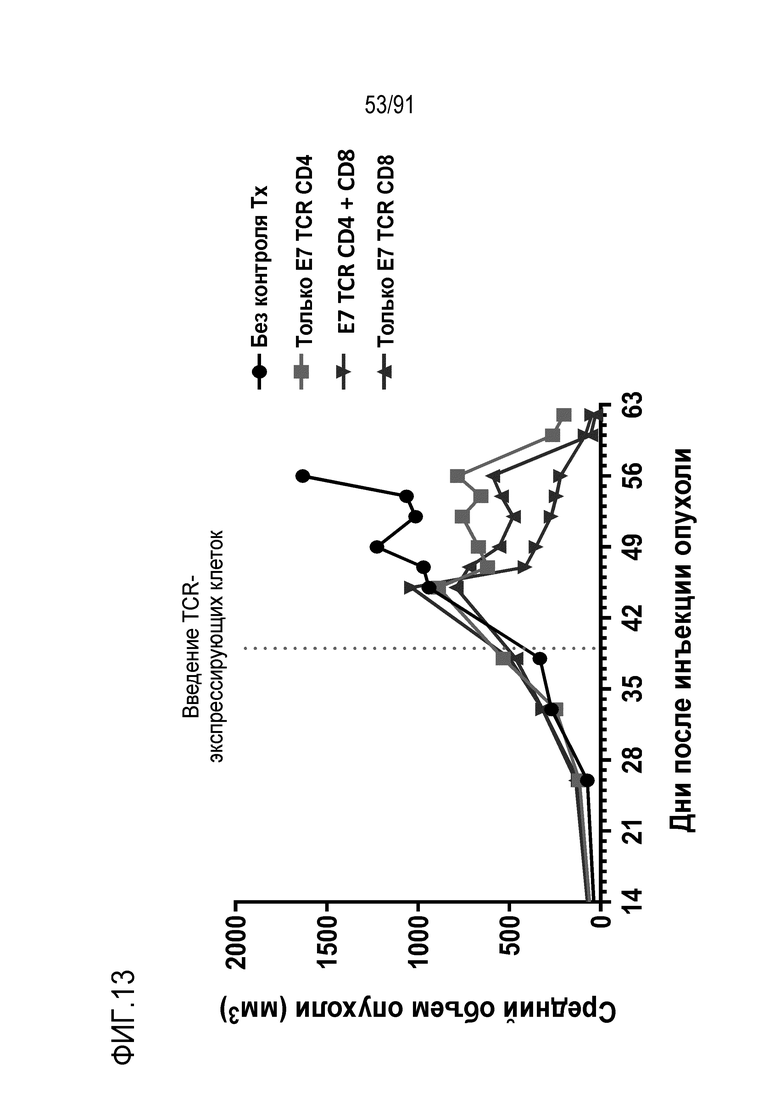
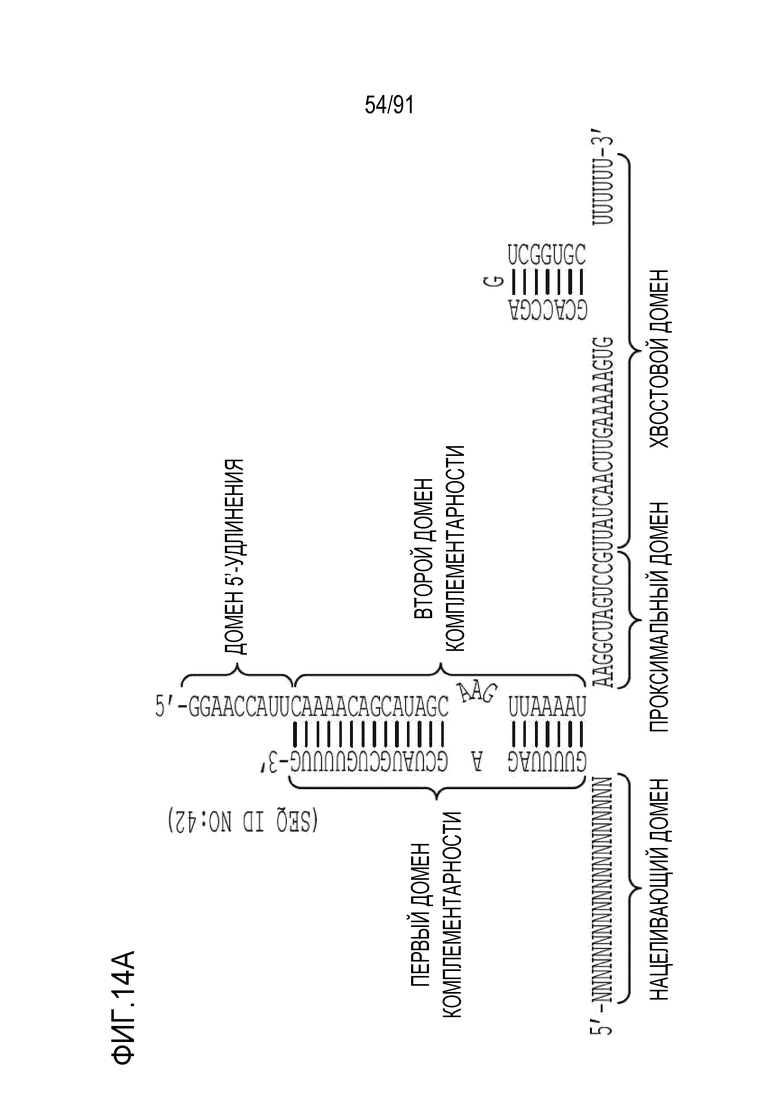
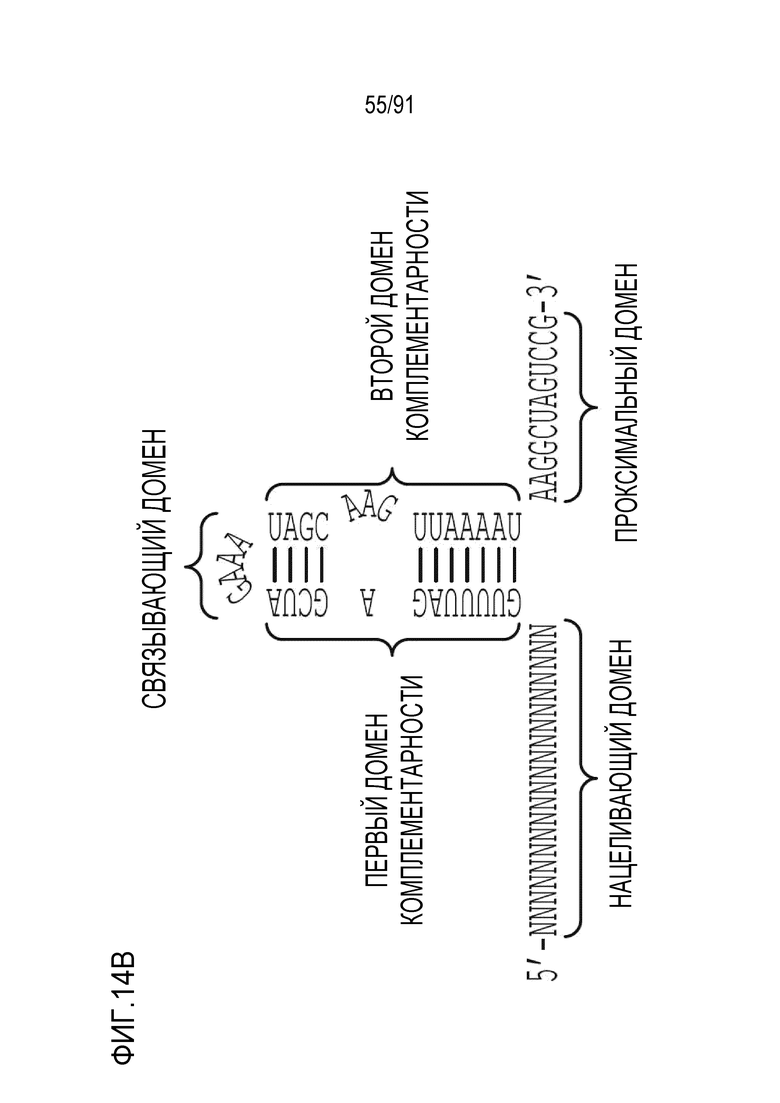
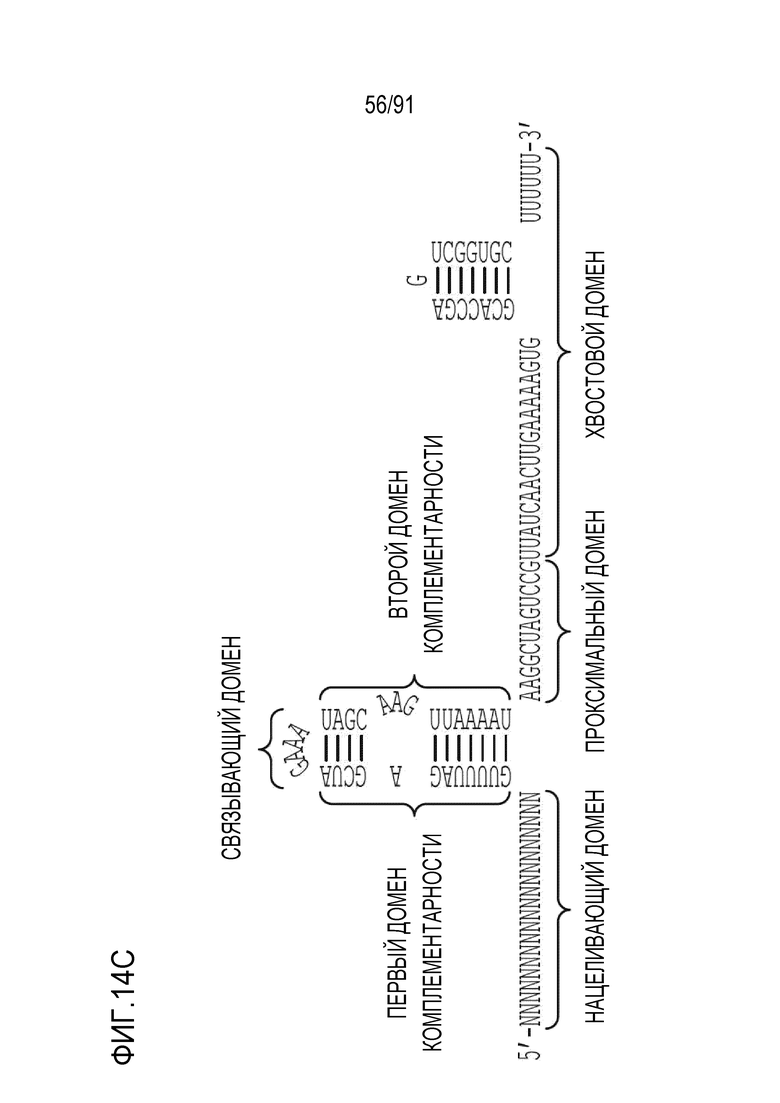
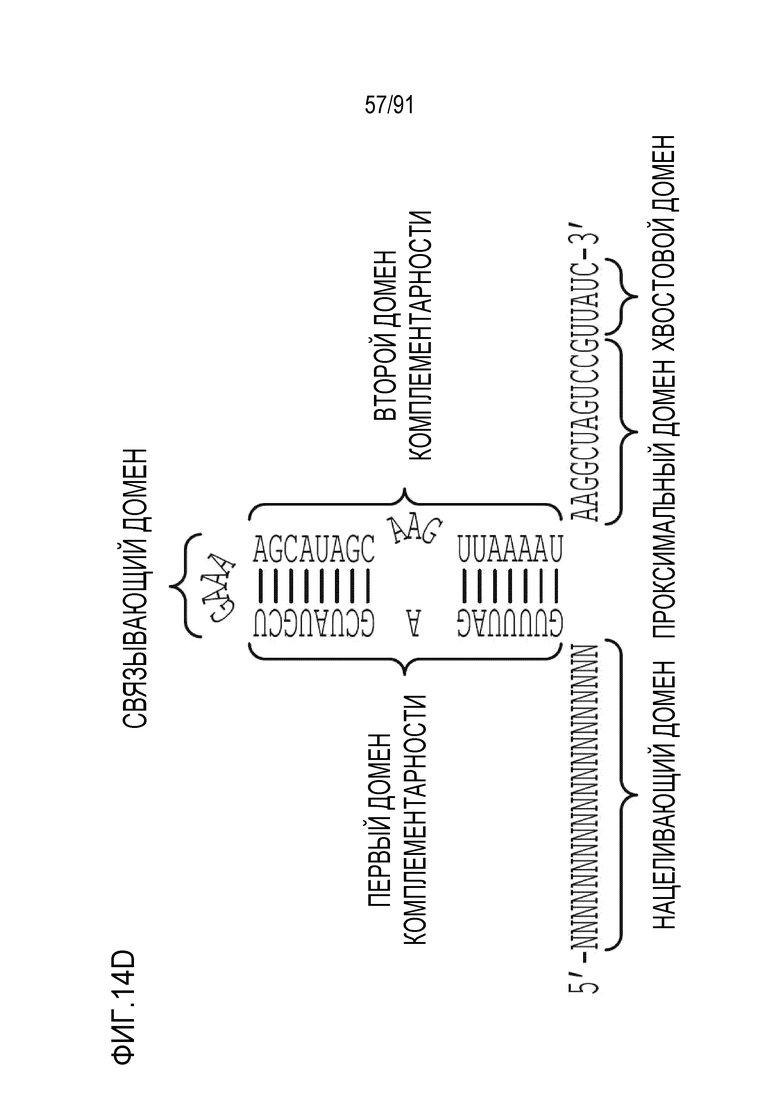
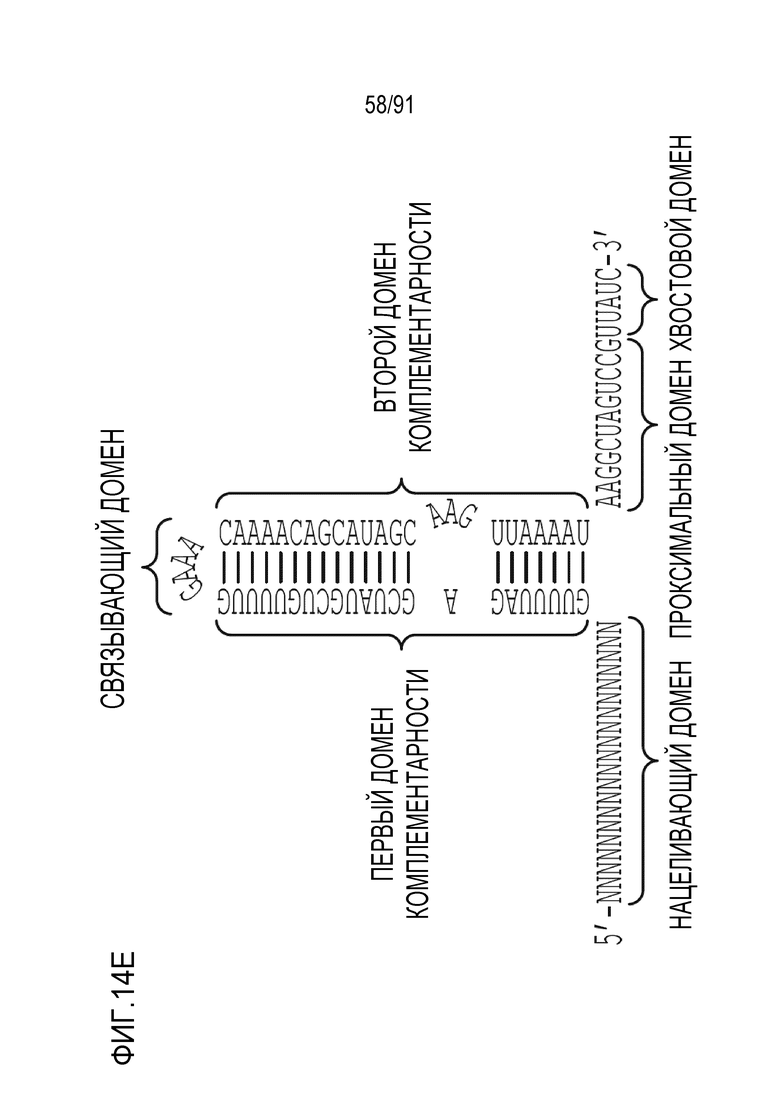
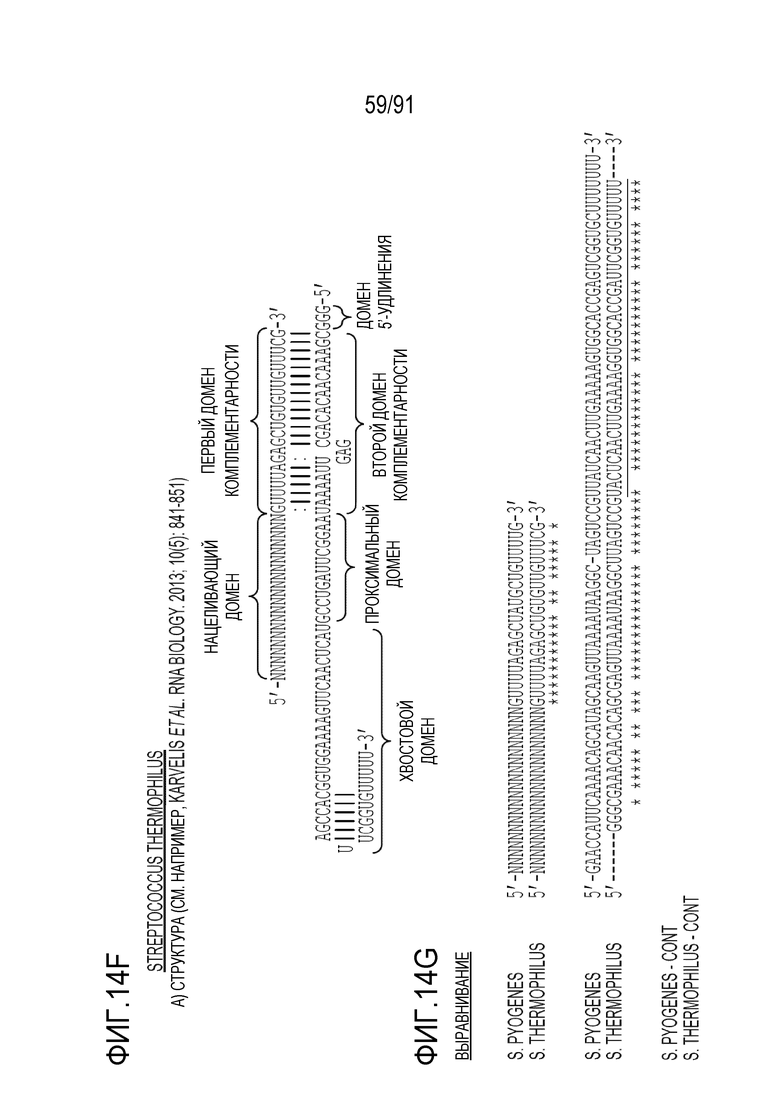
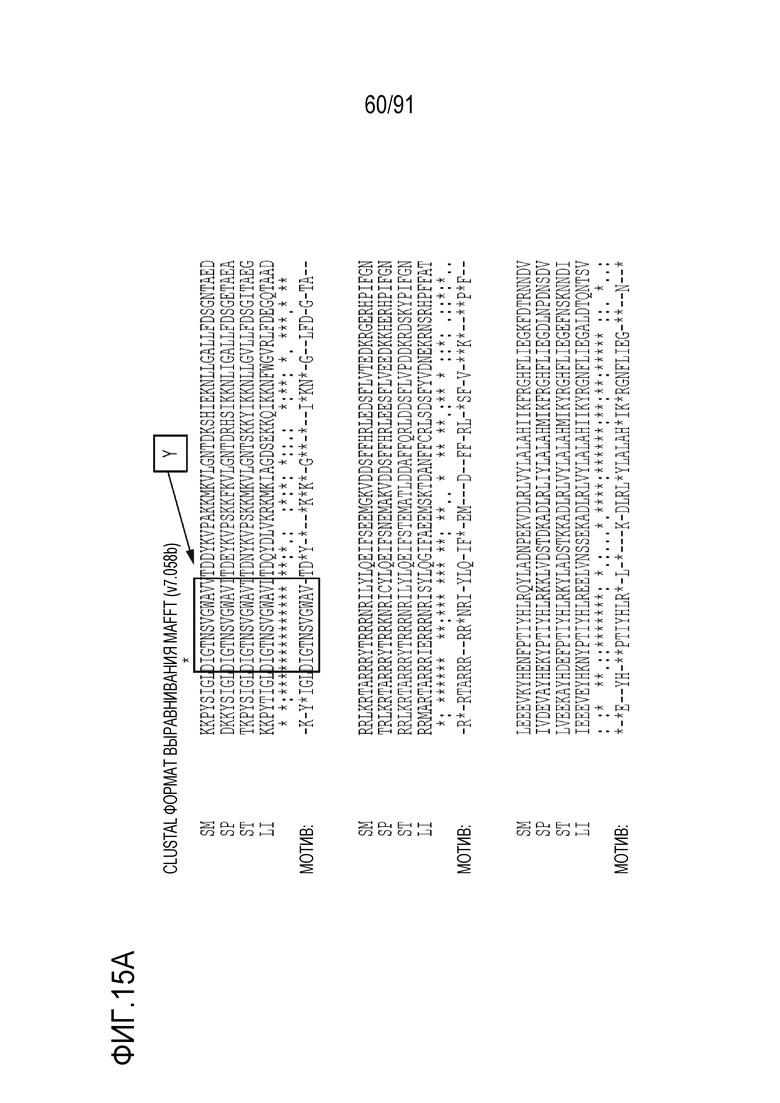
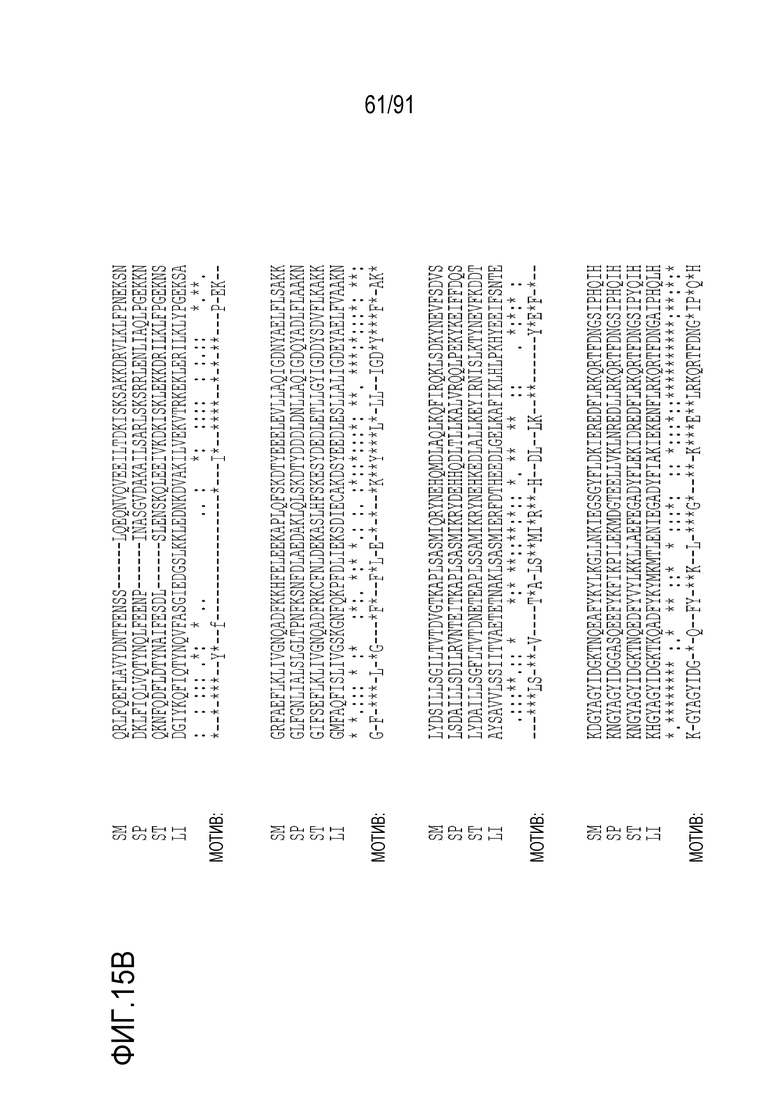

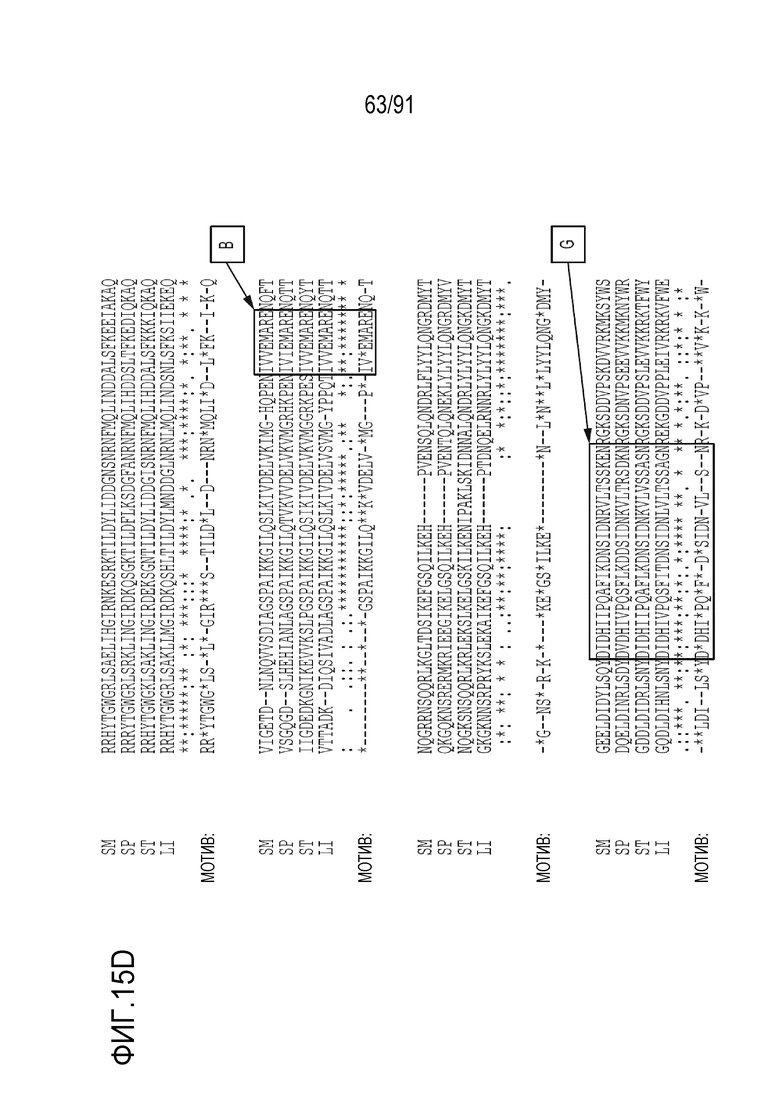
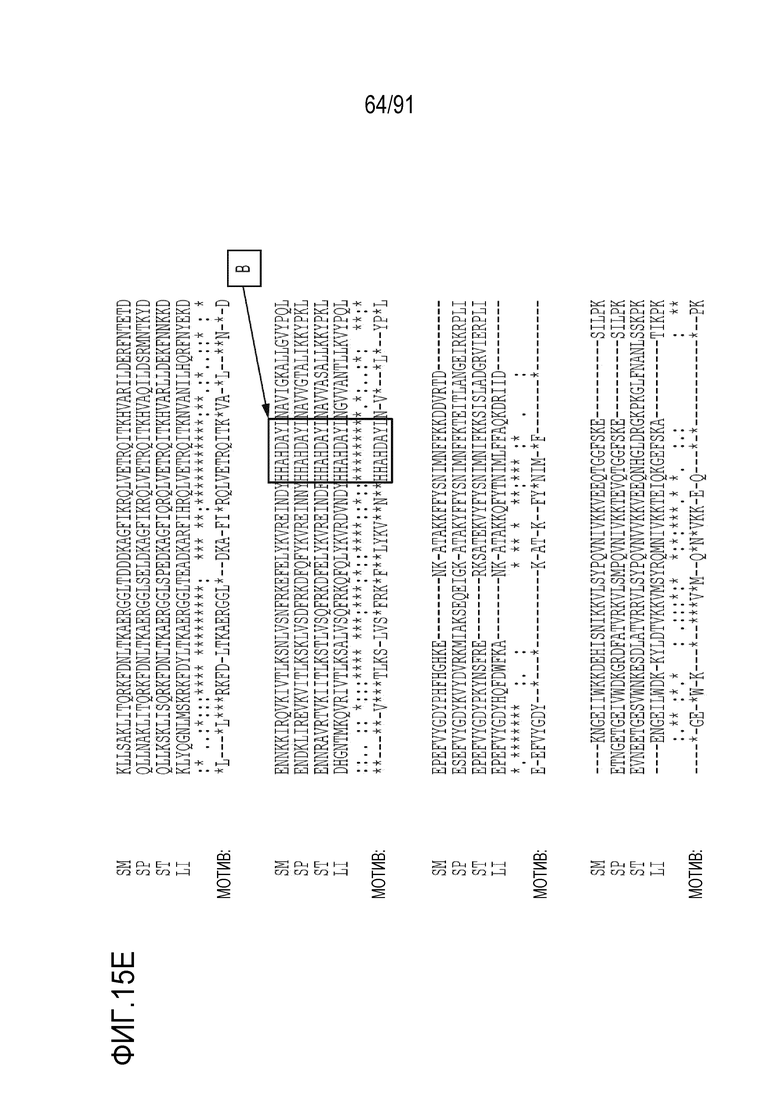





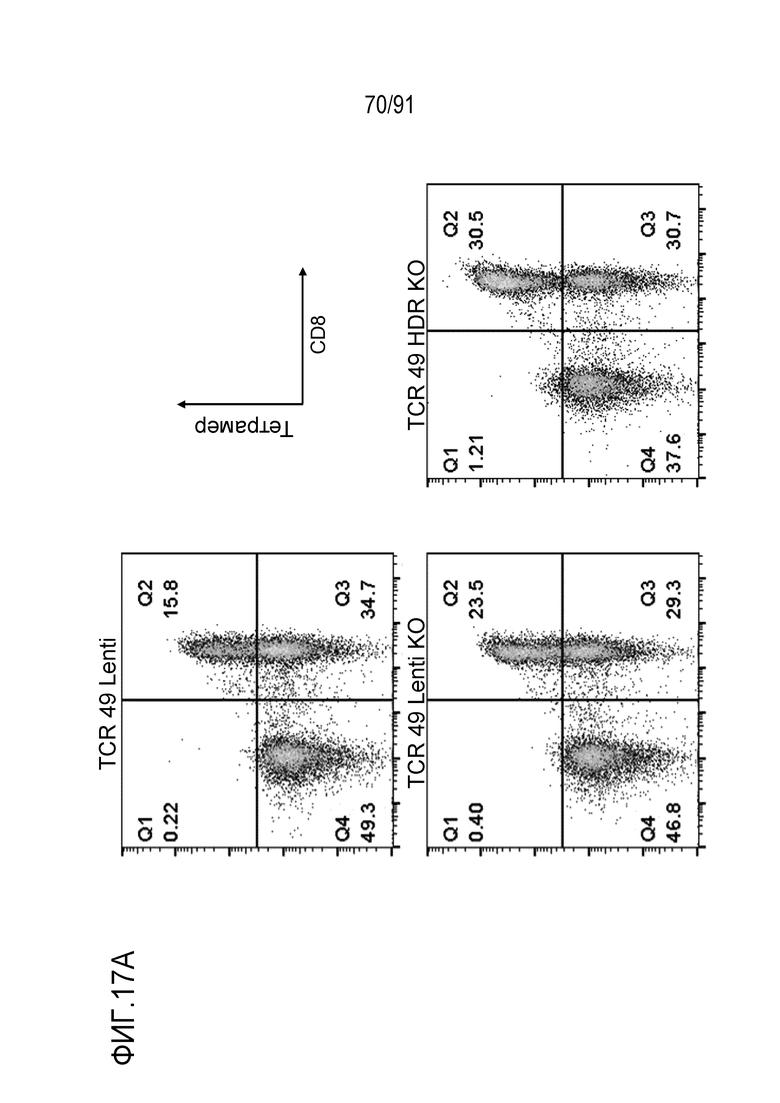
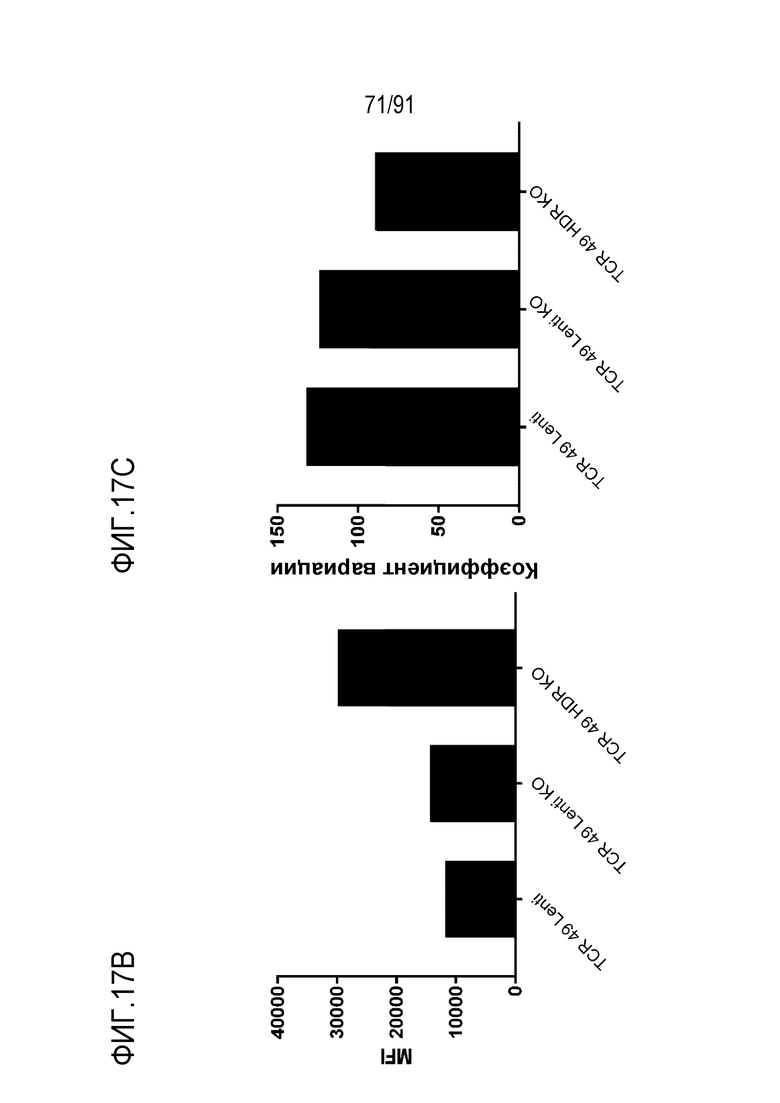
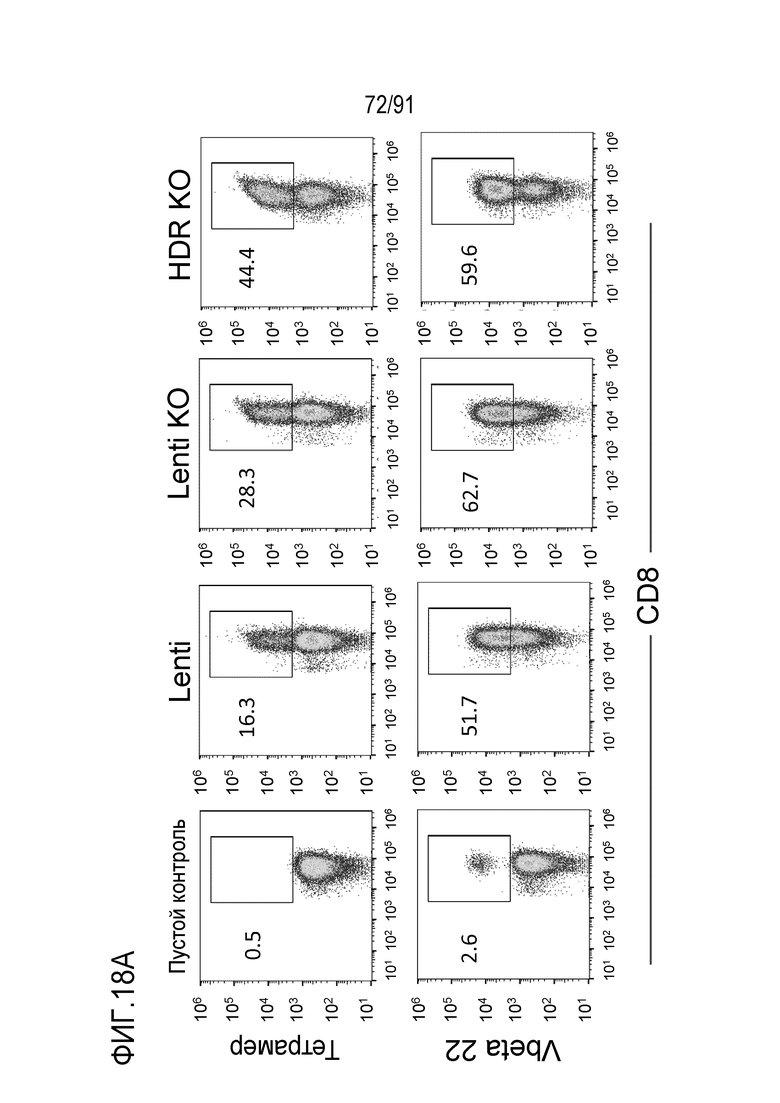
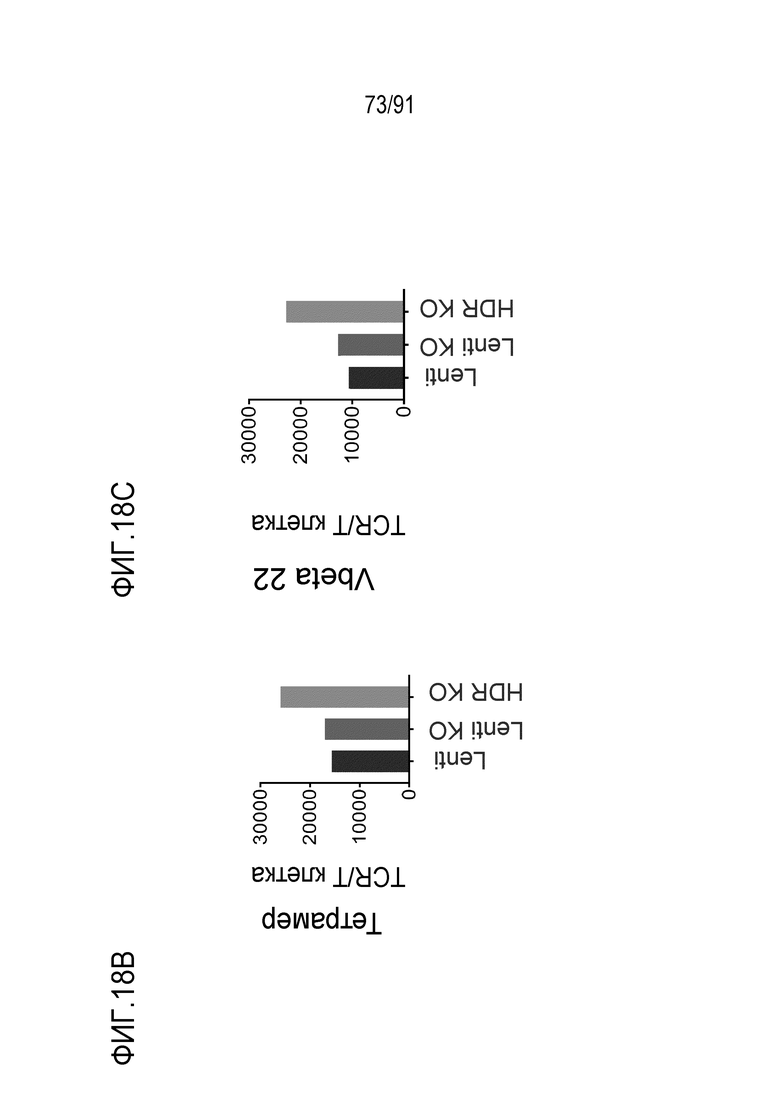
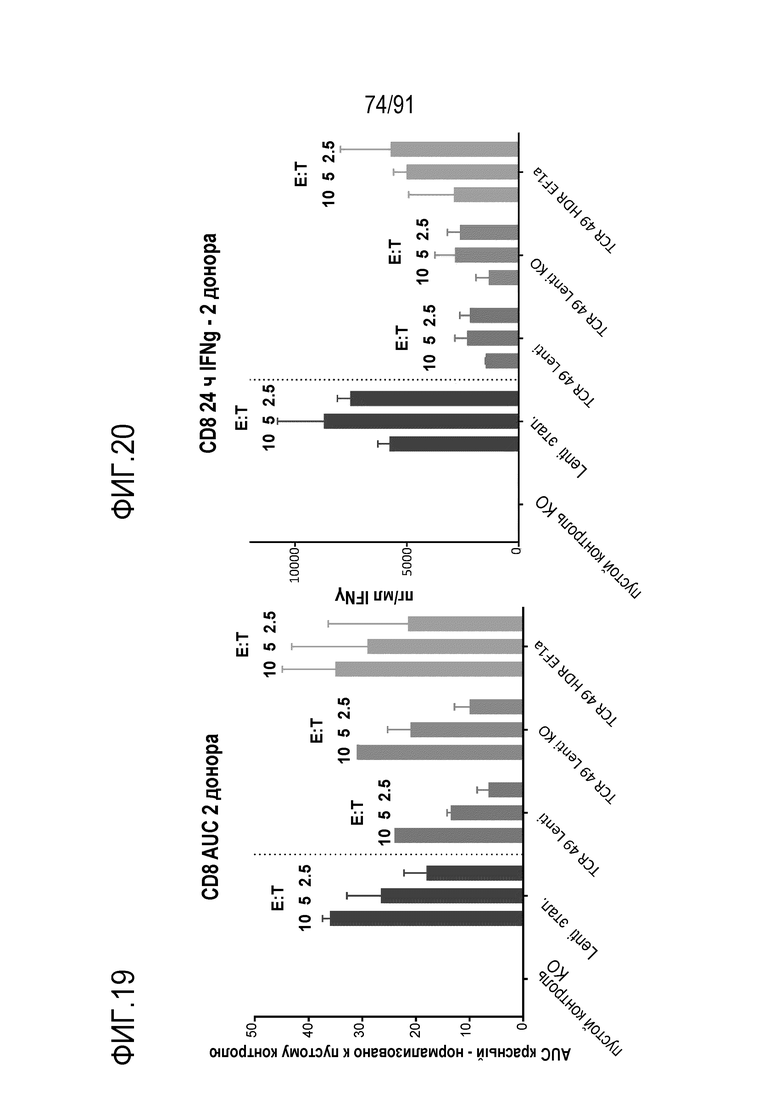
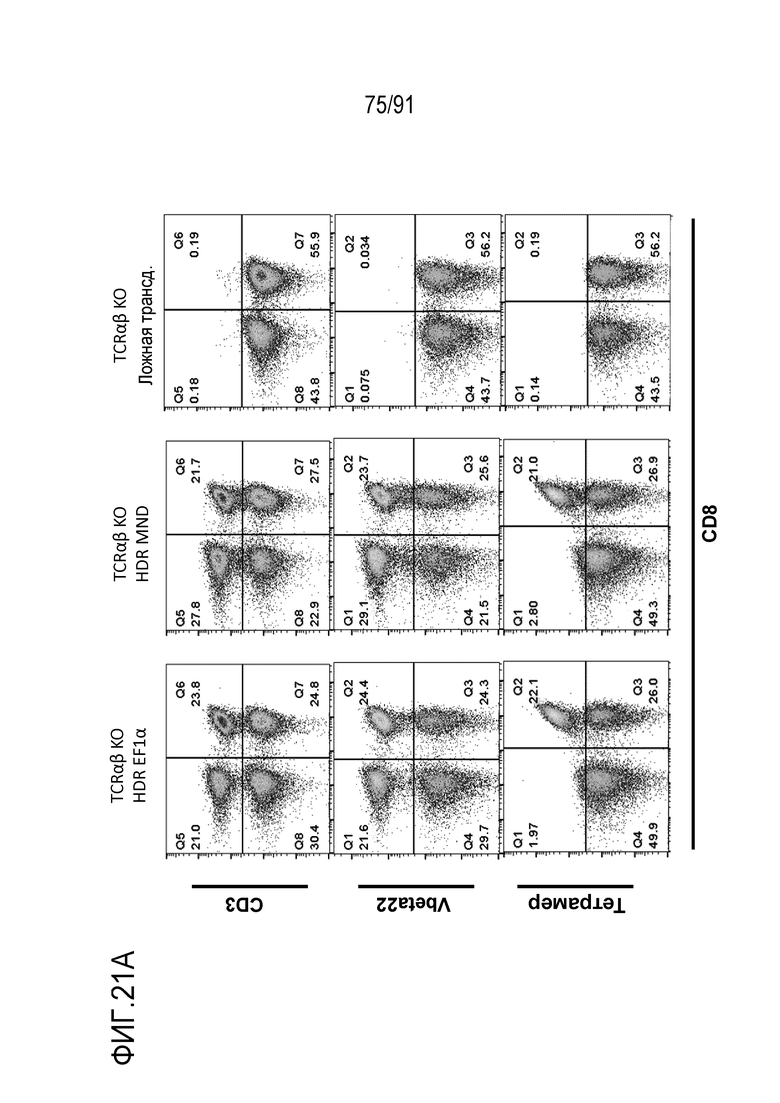
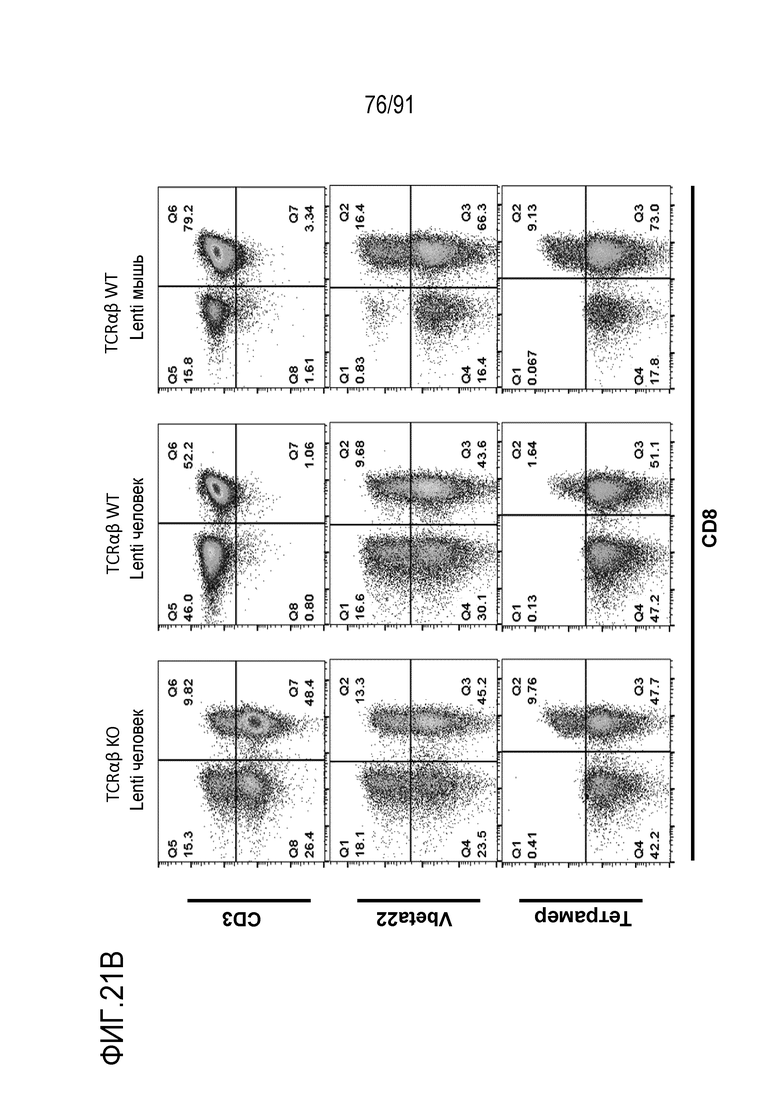
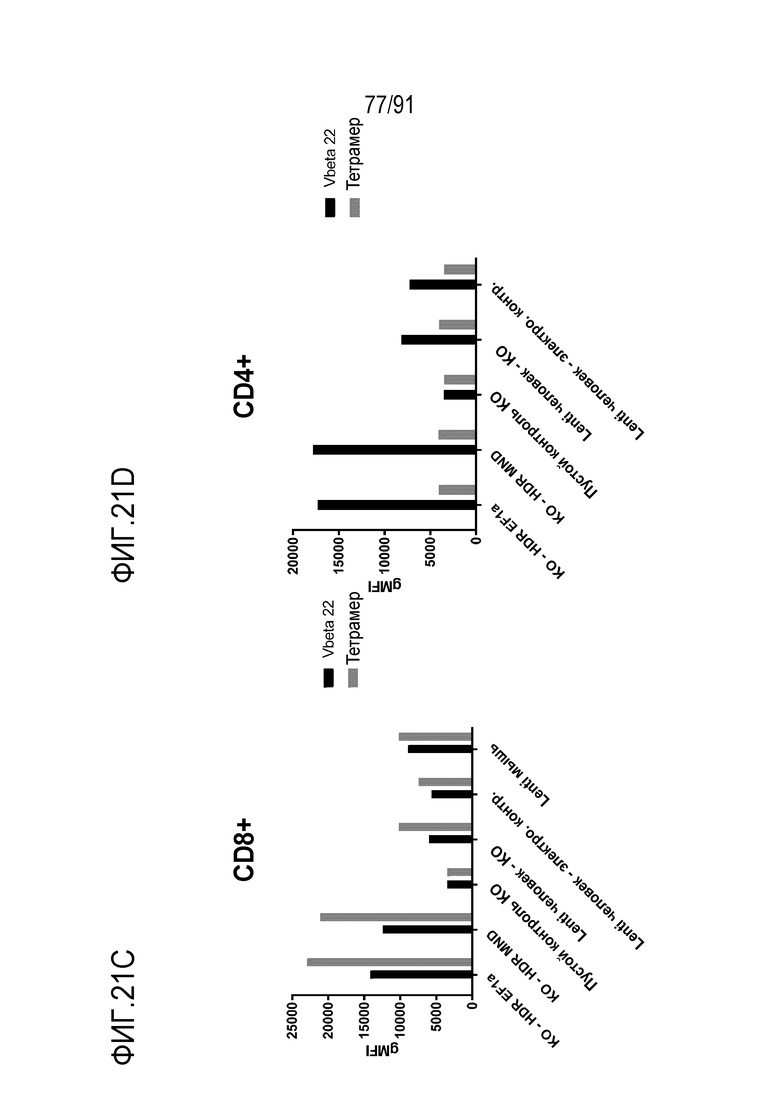
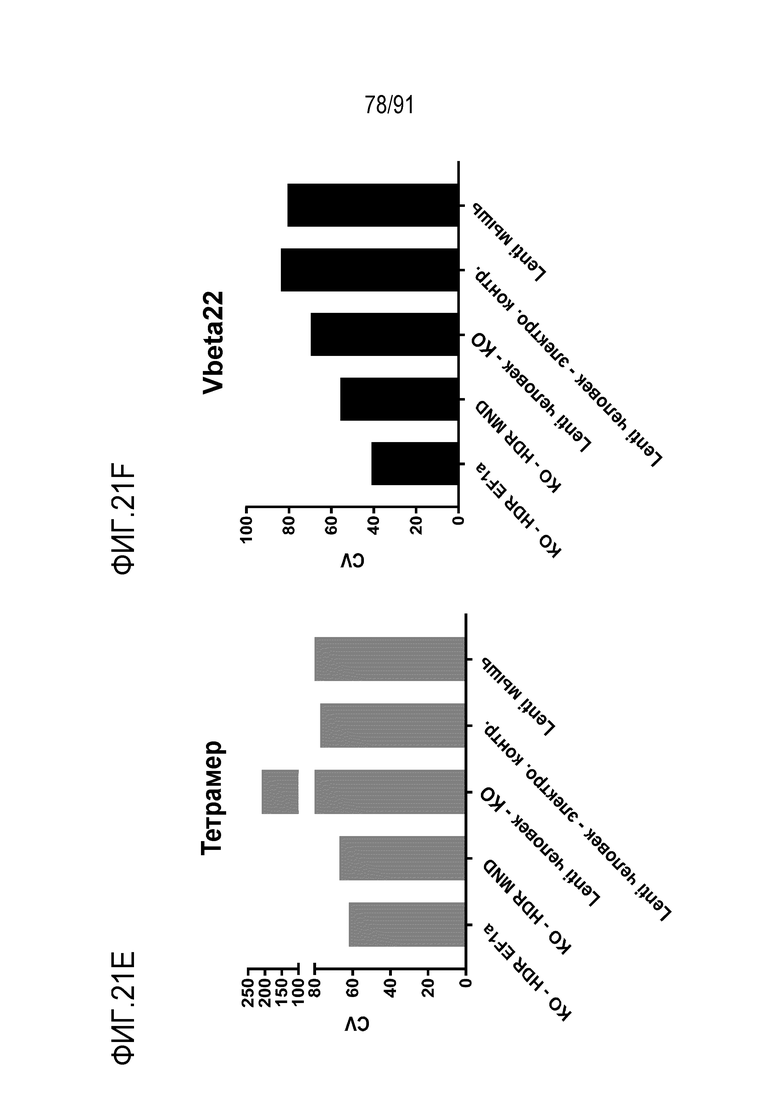
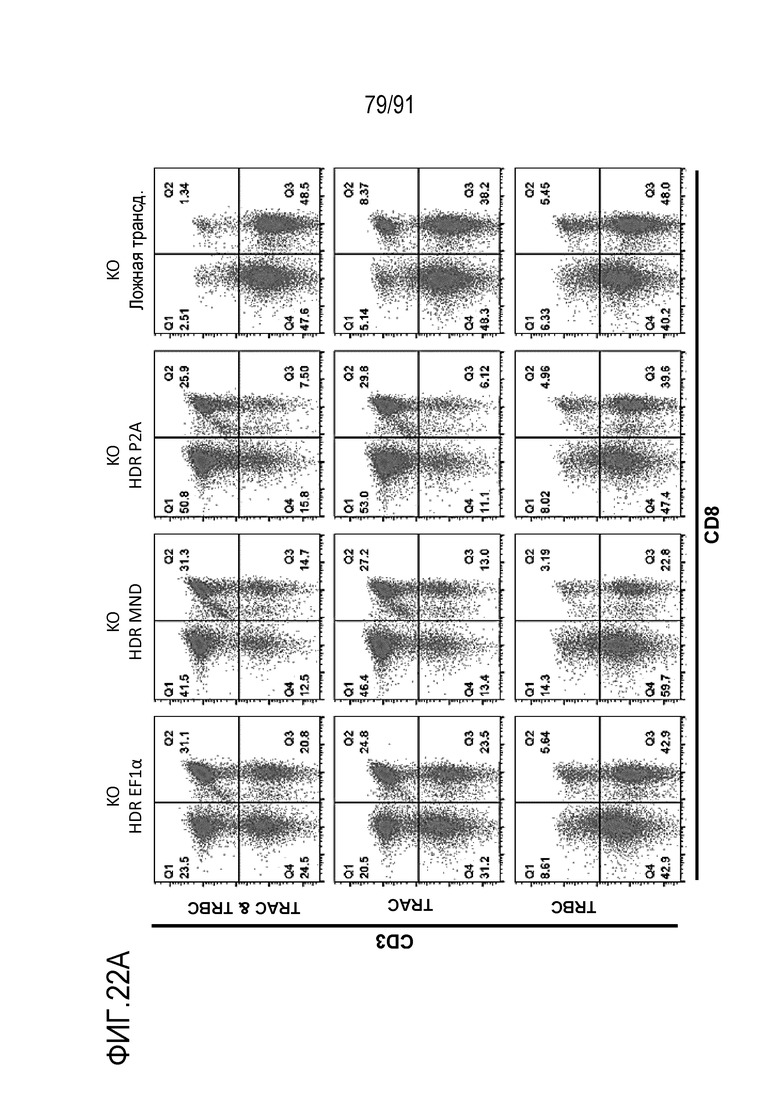
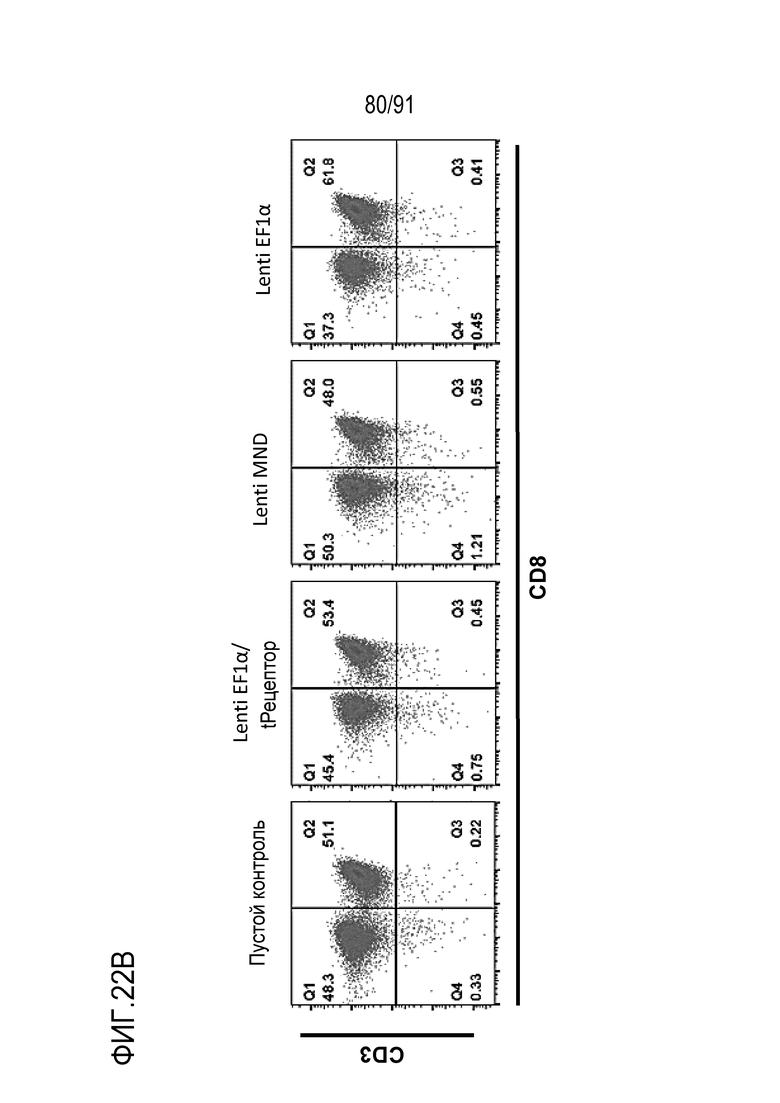
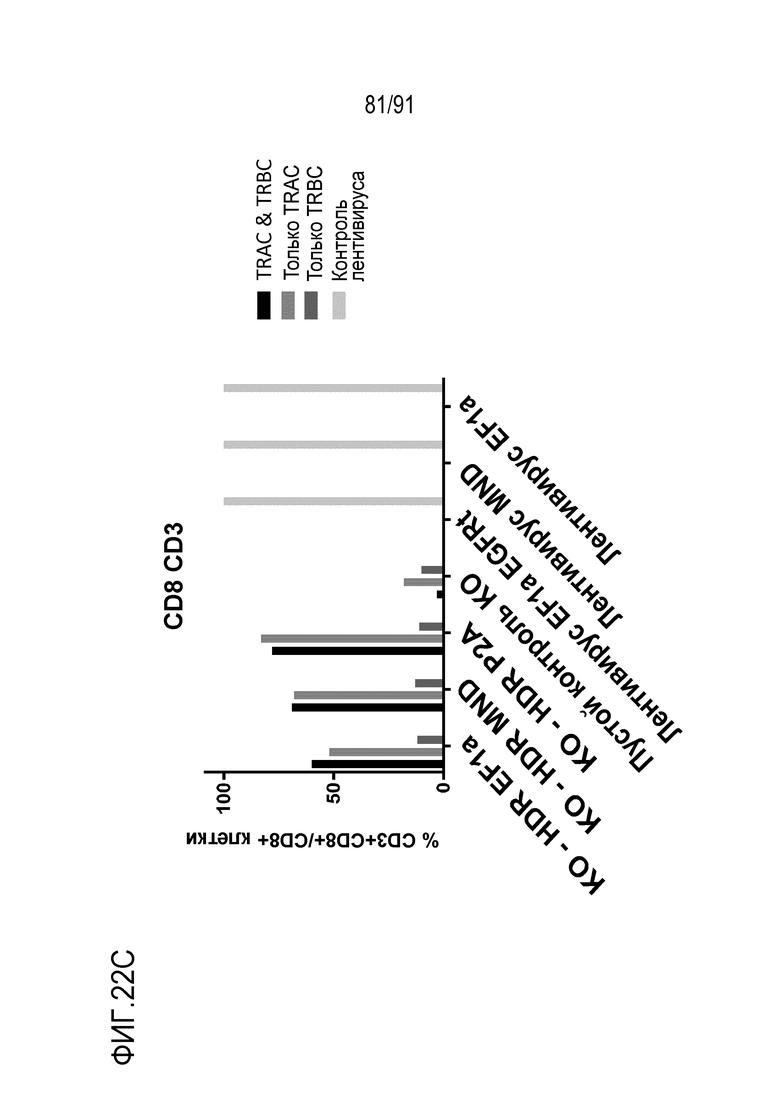

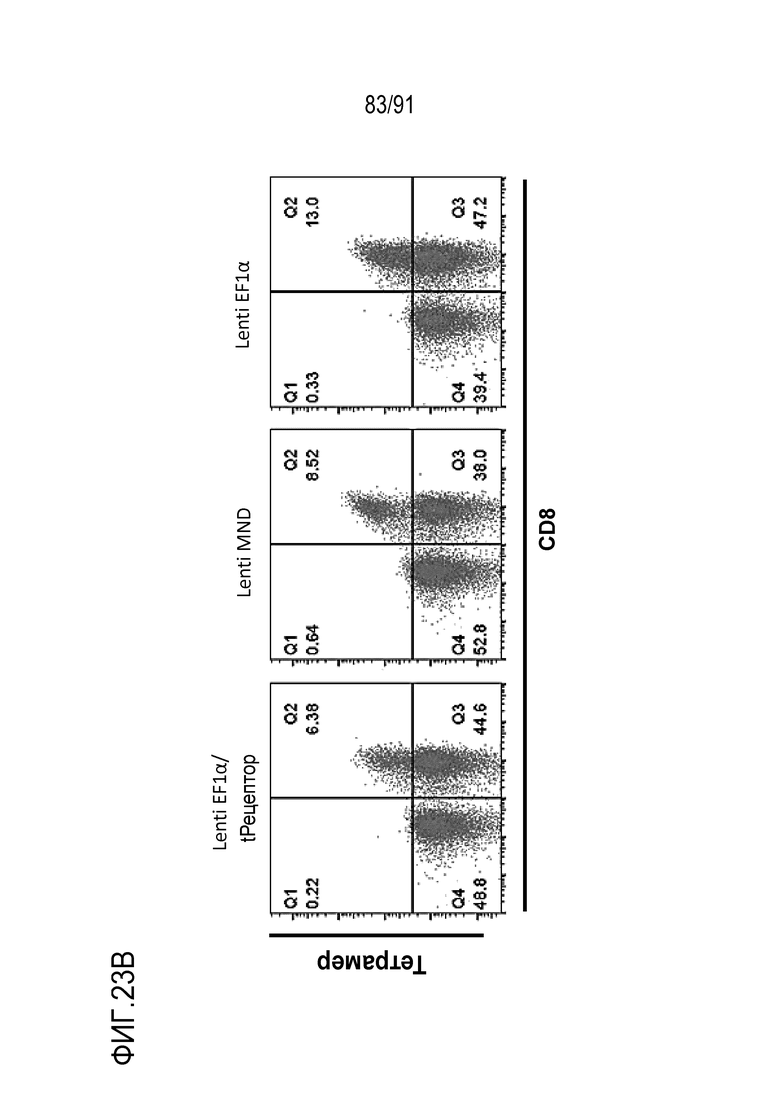
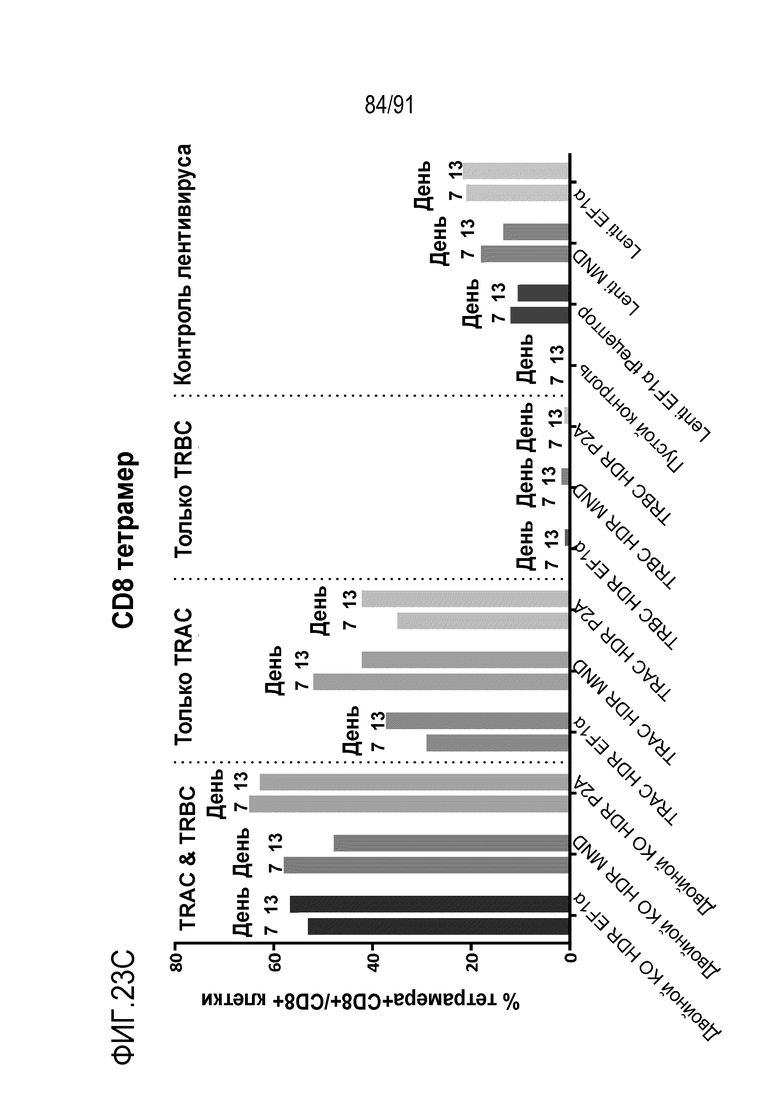
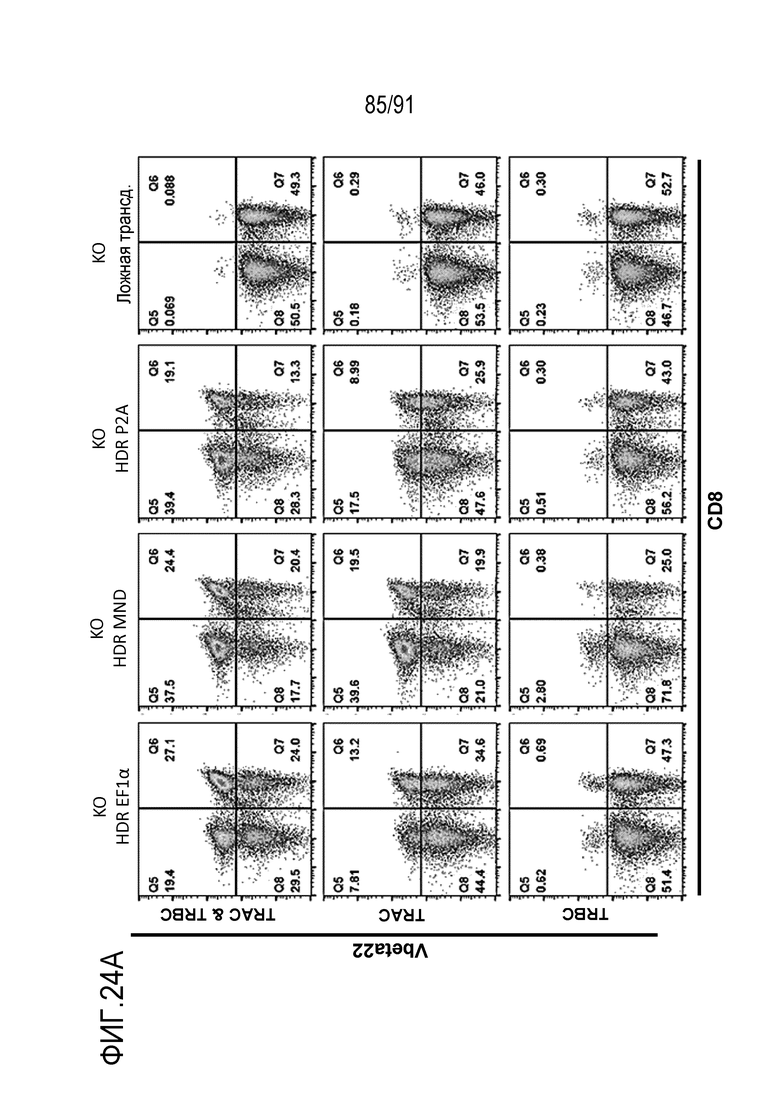
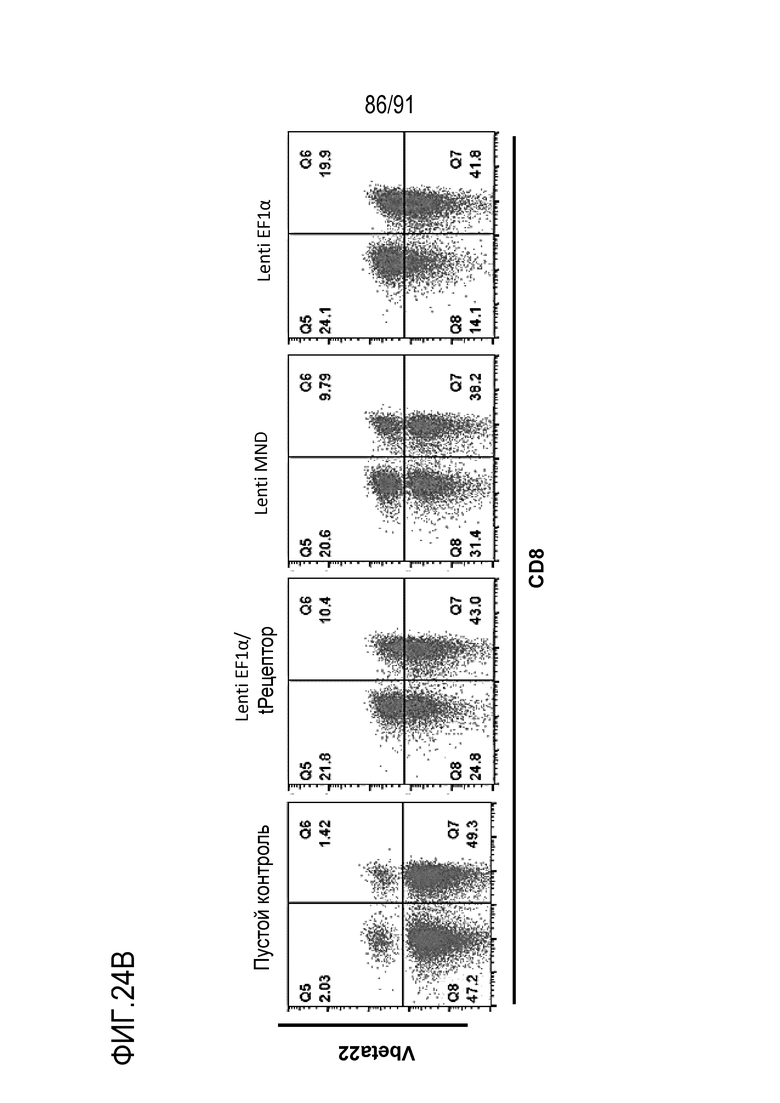
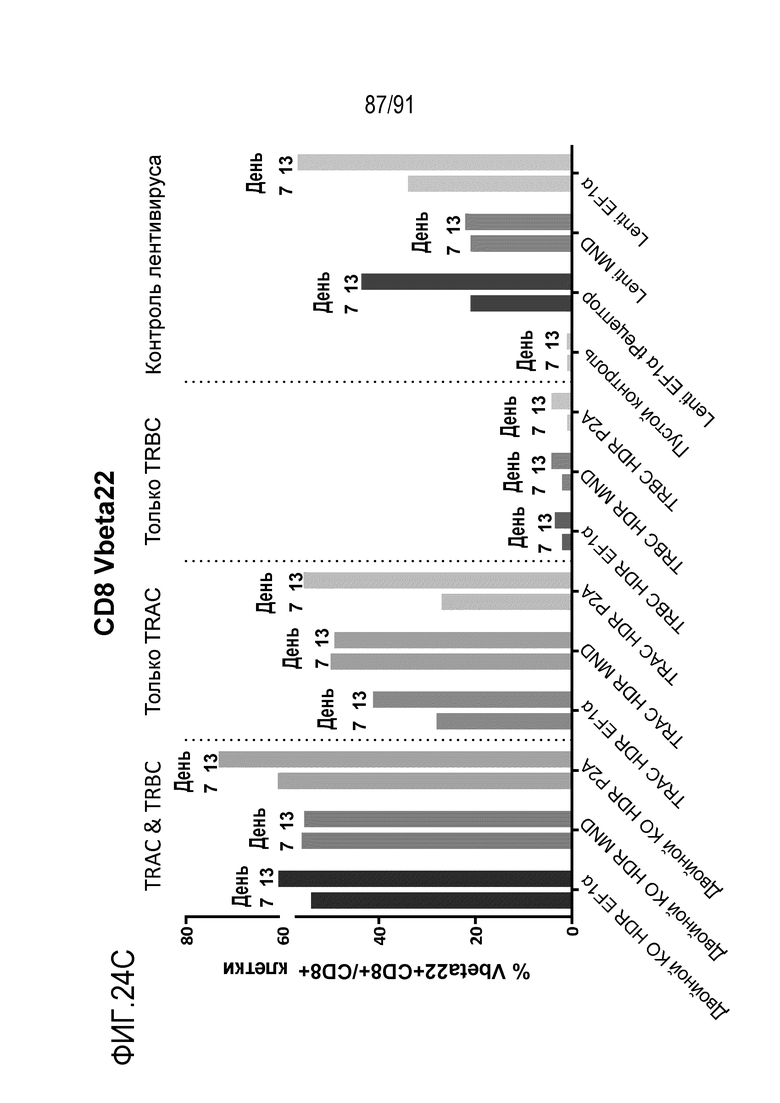
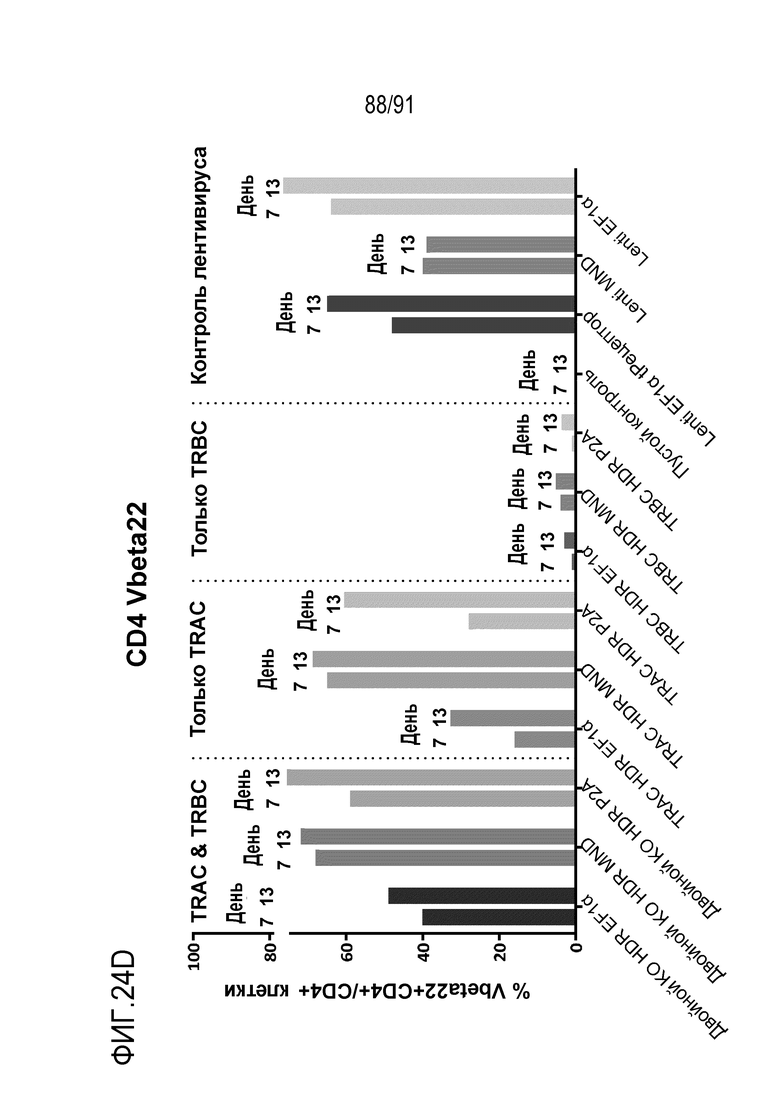
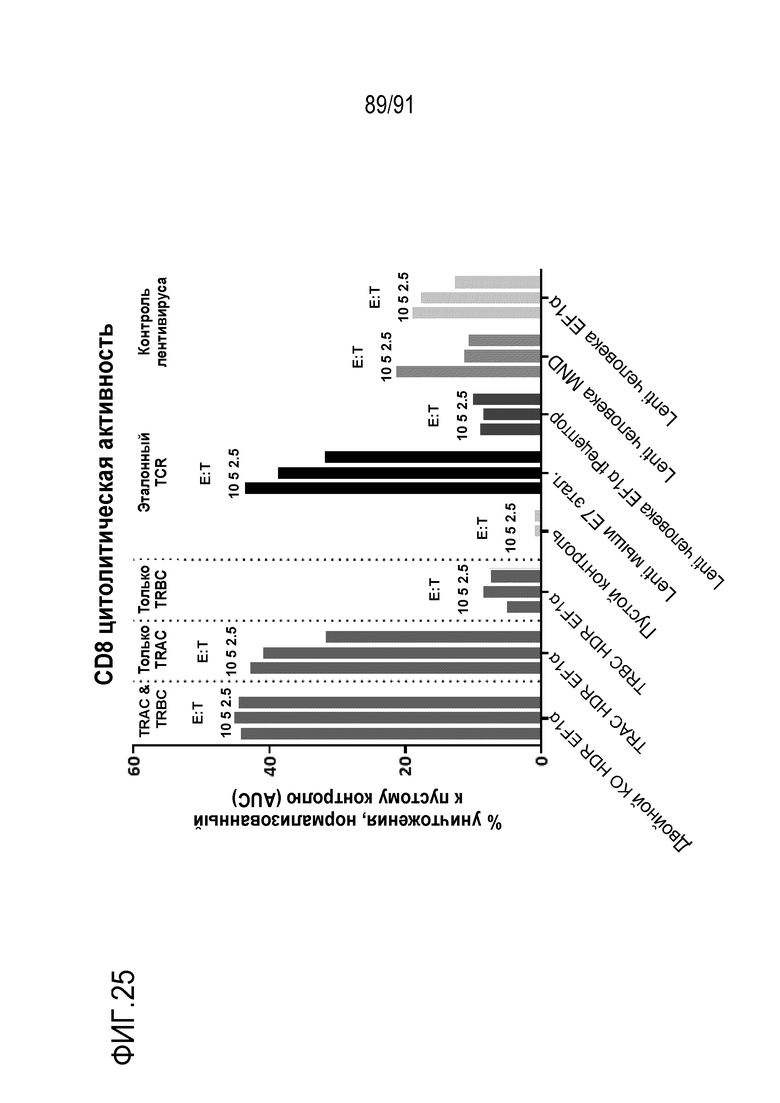
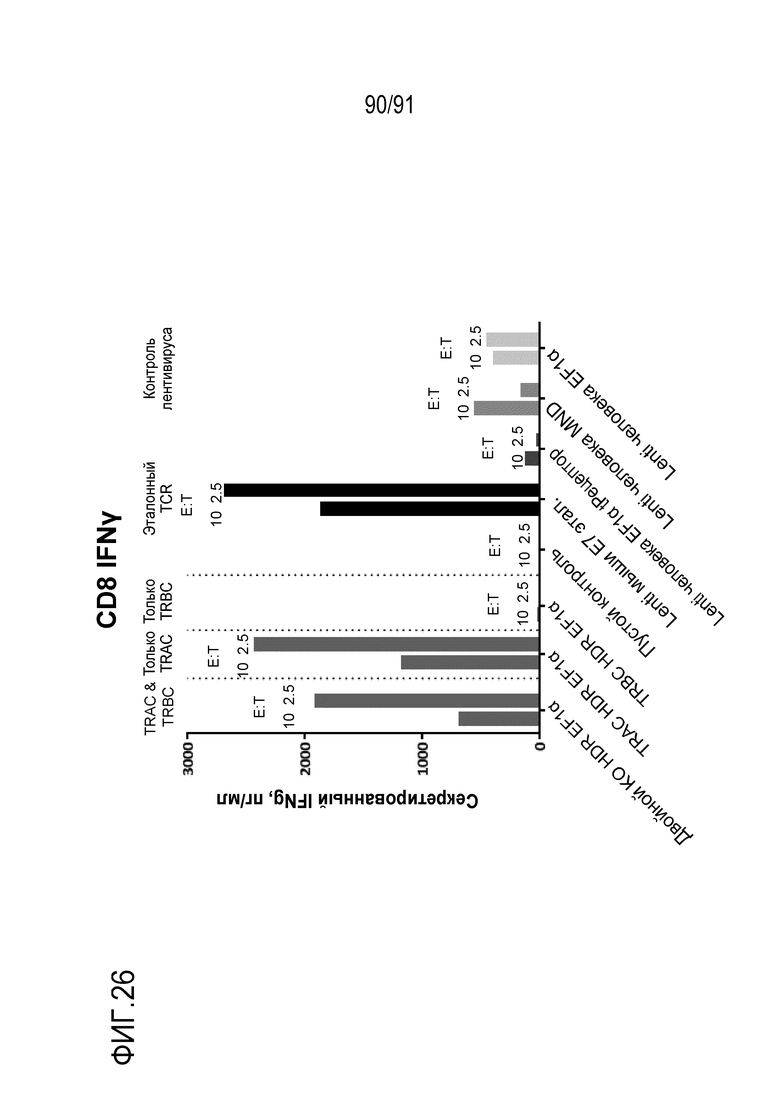

Изобретение относится к области биотехнологии, а именно к полинуклеотиду, кодирующему TCR для интеграции в локус константной области Т-клеточного рецептора альфа (TRAC) или локус константной области Т-клеточного рецептора бета (TRBC) посредством направляемой гомологией репарации, а также к содержащим его вектору и клетке. Также раскрыт способ получения клетки, содержащей вышеуказанный полинуклеотид. Изобретение также относится к композиции, содержащей клетку, включающую полинуклеотид, кодирующий TCR для интеграции в локус константной области Т-клеточного рецептора альфа (TRAC) или локус константной области Т-клеточного рецептора бета (TRBC). Изобретение эффективно для лечения заболевания или расстройства, ассоциированных с HPV16. 12 н. и 136 з.п. ф-лы, 27 ил., 5 табл., 14 пр.
1. Полинуклеотид, кодирующий TCR для интеграции в локус константной области Т-клеточного рецептора альфа (TRAC) или локус константной области Т-клеточного рецептора бета (TRBC) посредством направляемой гомологией репарации, где указанный полинуклеотид содержит:
(а) последовательность нуклеиновой кислоты, кодирующую TCR или его антигенсвязывающую часть, которые связываются или распознают пептидный эпитоп вируса папилломы человека (HPV) 16 в контексте молекулы МНС, и
(b) одно или более плеч гомологии, связанных с последовательностью нуклеиновой кислоты, где одно или более плеч гомологии содержат последовательность, гомологичную одной или более областям открытой рамки считывания локуса константной области Т-клеточного рецептора альфа (TRAC) или локуса константной области Т-клеточного рецептора бета (TRBC).
2. Полинуклеотид по п.1, где пептидный эпитоп представляет собой или содержит аминокислоты 11-19 E7 YMLDLQPET (SEQ ID NO:236).
3. Полинуклеотид по п.1, где пептидный эпитоп представляет собой или содержит аминокислоты 29-38 E6 TIHDIILECV (SEQ ID NO:233).
4. Полинуклеотид по любому из пп.1-3, где указанные одно или более плеч гомологии содержат последовательность, гомологичную одной или более областям открытой рамки считывания локуса TRAC.
5. Полинуклеотид по п.4, где указанные одно или более плеч гомологии содержат 5'-плечо гомологии и/или 3'-плечо гомологии, где:
i) 5'-плечо гомологии содержит:
1) последовательность, содержащую, или по меньшей мере, содержащую или по меньшей мере 150, 200, 250, 300, 350, 400, 450, 500, 550 или 600 смежных нуклеотидов последовательности, которая имеет по меньшей мере 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичности последовательности с последовательностью, представленной в SEQ ID NO: 1343;
2) последовательность, содержащую, или по меньшей мере, содержащую или, по меньшей мере 150, 200, 250, 300, 350, 400, 450, 500, 550 или 600 смежных нуклеотидов последовательности, представленной в SEQ ID NO: 1343; или
3) последовательность, представленную в SEQ ID NO: 1343, и/или
ii) 3'-плечо гомологии содержит:
1) последовательность, содержащую или по меньшей мере содержащую или по меньшей мере 150, 200, 250, 300, 350, 400, 450, 500, 550 или 600 смежных нуклеотидов последовательности, которая имеет, по меньшей мере, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичности последовательности с последовательностью, представленной в SEQ ID NO: 1344;
2) последовательность, содержащую или по меньшей мере содержащую или по меньшей мере 150, 200, 250, 300, 350, 400, 450, 500, 550 или 600 смежных нуклеотидов последовательности, представленной в SEQ ID NO: 1344; или
3) последовательность, представленную в SEQ ID NO: 1344.
6. Полинуклеотид по п.1, где указанная последовательность нуклеиновой кислоты, кодирующая TCR или его антигенсвязывающую часть, кодирует (i) бета-цепь Т-клеточного рецептора (TCRβ), содержащую вариабельную бета область (Vβ) указанных TCR или его антигенсвязывающей части и константную область бета (Cβ); и (ii) альфа-цепь Т-клеточного рецептора (TCRα), содержащую вариабельную область альфа (Vα) указанных TCR или его антигенсвязывающей части, где указанная цепь TCRα содержит полноразмерную цепь TCRα.
7. Полинуклеотид по п.1, где указанная последовательность нуклеиновой кислоты, кодирующая TCR или его антигенсвязывающую часть, кодирует (i) бета-цепь Т-клеточного рецептора (TCRβ), содержащую вариабельную бета область (Vβ) указанных TCR или его антигенсвязывающей части и константную область бета (Cβ); и (ii) часть альфа-цепи Т-клеточного рецептора (TCRα), содержащую вариабельную область альфа (Vα) указанных TCR или его антигенсвязывающей части, где указанная часть цепи TCRα представляет собой менее чем полноразмерную цепь TCRα.
8. Полинуклеотид по п.7, где цепь TCRα содержит константную область альфа (Cα), где по меньшей мере часть указанной Cα кодируется открытой рамкой считывания эндогенного локуса TRAC или его частичной последовательностью, когда TCR или его антигенсвязывающий фрагмент экспрессируется клеткой, в которую был введен полинуклеотид.
9. Полинуклеотид по п.7 или 8, где последовательность нуклеиновой кислоты (а) и одно из одного или более плеч гомологии вместе содержат последовательность нуклеотидов, кодирующую Сα, которая меньше, чем полная длина нативной Сα, где по меньшей мере часть Cα кодируется открытой рамкой считывания эндогенного локуса TRAC или его частичной последовательностью, когда TCR или его антигенсвязывающий фрагмент экспрессируется клеткой, в которую был введен полинуклеотид.
10. Полинуклеотид по п.6, где последовательность нуклеиновой кислоты, кодирующая цепь TCRβ, находится в 5'-области относительно последовательности нуклеиновой кислоты, кодирующей цепь TCRα или часть цепи TCRα.
11. Полинуклеотид по п.6, где последовательность нуклеиновой кислоты, кодирующая TCR или его антигенсвязывающую часть, не содержит интрона.
12. Полинуклеотид по п.6, где последовательность нуклеиновой кислоты, кодирующая TCR или его антигенсвязывающую часть, представляет собой последовательность, которая является экзогенной или гетерологичной относительно открытой рамки считывания эндогенного геномного локуса TRAC Т-клетки, необязательно человеческой Т-клетки.
13. Полинуклеотид по п.6, где последовательность нуклеиновой кислоты, кодирующая TCR или его антигенсвязывающую часть, находится в рамке с одним или более экзонами или их частичной последовательностью.
14. Полинуклеотид по п.8 или 9, где Cα кодируется открытой рамкой считывания эндогенного локуса TRAC или его частичной последовательностью, и где дополнительная часть Cα кодируется последовательностью нуклеиновой кислоты, кодирующей TCR или его антигенсвязывающую часть, где указанная дополнительная часть Cα меньше чем полная длина нативной Cα.
15. Полинуклеотид по п.14, где указанная дополнительная часть Cα кодируется последовательностью нуклеотидов, начиная с остатка 3 и до остатка 3155 последовательности, представленной в SEQ ID NO: 348, или одним или более ее экзонами или последовательностью, которая имеет по меньшей мере 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичности последовательности с последовательностью нуклеотидов, начиная с остатка 3 и до остатка 3155 последовательности, представленной в SEQ ID NO: 348, или с одним или более ее экзонами, или их частичной последовательностью.
16. Полинуклеотид по п.14 или 15, где указанная дополнительная часть Cα кодируется последовательностью, представленной в SEQ ID NO: 1364, или последовательностью, которая имеет по меньшей мере 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичности последовательности с SEQ ID NO: 1364 или ее частичной последовательностью.
17. Полинуклеотид по п.14, где указанная дополнительная часть области Cα и/или области Cβ, кодируемой последовательностью нуклеиновой кислоты (а), содержит одну или более модификаций, необязательно замену, делецию или вставку одной или более аминокислот по сравнению с нативной областью Cα и/или нативной областью Cβ, необязательно указанные одна или более модификаций вводят один или более остатков цистеина, которые способны образовывать одну или более ненативных дисульфидных связей между альфа-цепью и бета-цепью.
18. Полинуклеотид по п.6, где последовательность нуклеиновой кислоты, кодирующая TCR или его антигенсвязывающую часть, содержит один или более полицистронных элементов.
19. Полинуклеотид по п.18, где полистронный элемент(элементы) расположен между последовательностью нуклеиновой кислоты, кодирующей TCRα или ее часть, и последовательностью нуклеиновой кислоты, кодирующей TCRβ или ее часть.
20. Полинуклеотид по п.18 или 19, где один или более полицистронных элементов находятся в 5'-области относительно последовательности нуклеиновой кислоты, кодирующей TCR, или часть TCR, или молекулы нуклеиновой кислоты, кодирующей TCR.
21. Полинуклеотид по п.18, где один или более полицистронных элементов представляют собой или содержат последовательность пропуска рибосомы, где последовательность пропуска рибосомы представляет собой элемент T2A, P2A, E2A или F2A.
22. Полинуклеотид по п.6, где указанный полинуклеотид соединен с одним или более гетерологичных или регуляторных контрольных элементов, функционально связанных с контролем экспрессии TCR, когда он экспрессируется клеткой, в которую был введен полинуклеотид.
23. Полинуклеотид по п.22, где один или более гетерологичных регуляторных или контрольных элементов содержат промотор, энхансер, интрон, сигнал полиаденилирования, консенсусную последовательность Козака, акцепторную последовательность сплайсинга и/или донорную последовательность сплайсинга.
24. Полинуклеотид по п.22 или 23, где гетерологичный регуляторный или контрольный элемент содержат гетерологичный промотор, необязательно промотор фактора элонгации 1 альфа (EF1α) человека или промотор MND, или его вариант.
25. Полинуклеотид по п. 1, который представляет собой линейный полинуклеотид.
26. Вектор для доставки полинуклеотида, содержащий полинуклеотид по п.1.
27. Вектор по п.26, где вектор представляет собой экспрессирующий вектор.
28. Вектор по п.26 или 27, где вектор представляет собой вирусный вектор.
29. Вектор по п.28, где вирусный вектор представляет собой ретровирусный вектор.
30. Вектор по п.28 или 29, где вирусный вектор представляет сбой лентивирусный вектор.
31. Вектор по п.30, где лентивирусный вектор получен из HIV-1.
32. Вектор по п.28 или 29, где вирусный вектор представляет собой гаммаретровирусный вектор.
33. Вектор по п.28, где вирусный вектор представляет собой вектор AAV.
34. Вектор по п. 33, где вектор AAV выбран из векторов AAV1, AAV2, AAV3, AAV4, AAV5, AAV6, AAV7 или AAV8.
35. Сконструированная клетка для экспрессии рекомбинантного Т-клеточного рецептора, интегрированного в локус константной области Т-клеточного рецептора альфа (TRAC) или локус константной области Т-клеточного рецептора бета (TRBC) клетки, содержащая полинуклеотид по любому из пп.1-25 или вектор по любому из пп.26-34, где указанная сконструированная клетка представляет собой T-клетку.
36. Сконструированная клетка по п.35, содержащая генетическое нарушение гена константной области эндогенного Т-клеточного рецептора альфа (TRAC) и/или гена константной области Т-клеточного рецептора бета (TRBC).
37. Сконструированная клетка по п.36, где ген TRBC представляет собой один или оба из гена константной области 1 Т-клеточного рецептора бета (TRBC1) или гена константной области 2 Т-клеточного рецептора бета (TRBC2).
38. Сконструированная клетка по п.36 или 37, где сконструированная клетка не содержит непрерывного гена TRAC и/или TRBC; не содержит ген TRAC и/или TRBC; не содержит функционального гена TRAC и/или TRBC; и/или не экспрессирует, не экспрессирует на детектируемом уровне или экспрессирует на уровне менее чем 20%, 15%, 10%, 5%, 4%, 3%, 2% или 1% от уровня генного продукта эндогенного TRAC или TRBC дикого типа.
39. Сконструированная клетка по п.38, где генный продукт представляет собой мРНК или белок, кодируемый геном TRAC или TRBC.
40. Сконструированная клетка по п.36, где генетическое нарушение включает мутацию или делецию в области гена TRAC или TRBC, которые находятся в кодирующей области, необязательно ранней кодирующей области гена, в пределах экзона 1 гена, в кодирующей области в пределах 500, 400, 300, 200, 100 или 50 пар оснований от старт-кодона гена, в последовательности сайта-мишени, которая комплементарна нацеливающему сайту нацеливающего домена гид-РНК (гРНК), имеющего последовательность, выбранную из любой из SEQ ID NO: 1053 и 1259-1315, и/или с которой специфически гибридизуется нацеливающий домен, имеющий последовательность, выбранную из SEQ ID NO: 1053 и 1259-1315, и/или в последовательности сайта-мишени, которая комплементарна нацеливающему сайту нацеливающего домена гРНК, имеющего последовательность, выбранную из любой из SEQ ID NO: 1048 и 1229-1258, и/или с которой специфически гибридизуется нацеливающий домен, имеющий последовательность, выбранную из SEQ ID NO: 1048 и 1229-1258.
41. Сконструированная клетка по п.36, где на генетическое нарушение влияет один или более агентов, которые содержат (a) по меньшей мере одну гРНК, имеющую нацеливающий домен, который комплементарен домену-мишени гена TRAC и/или гена TRBC или (b) по меньшей мере одну нуклеиновую кислоту, кодирующую по меньшей мере одну гРНК.
42. Сконструированная клетка по п.41, где один или более агентов содержат, по меньшей мере, один комплекс молекулы Cas9 и, по меньшей мере, одну гРНК, имеющую нацеливающий домен, который комплементарен домену-мишени гена TRAC и/или гена TRBC.
43. Сконструированная клетка по любому из пп.41 или 42, где по меньшей мере одна гРНК содержит нацеливающий домен, который комплементарен домену-мишени гена TRAC, причем указанный нацеливающий домен содержит последовательность, выбранную из UCUCUCAGCUGGUACACGGC (SEQ ID NO:1229), UGGAUUUAGAGUCUCUCAGC (SEQ ID NO:1230), ACACGGCAGGGUCAGGGUUC (SEQ ID NO:1231), GAGAAUCAAAAUCGGUGAAU (SEQ ID NO:1048), GCUGGUACACGGCAGGGUCA (SEQ ID NO:1232), CUCAGCUGGUACACGGC (SEQ ID NO:1233), UGGUACACGGCAGGGUC (SEQ ID NO:1234), GCUAGACAUGAGGUCUA (SEQ ID NO:1235), GUCAGAUUUGUUGCUCC (SEQ ID NO:1236), UCAGCUGGUACACGGCA (SEQ ID NO:1237), GCAGACAGACUUGUCAC (SEQ ID NO:1238), GGUACACGGCAGGGUCA (SEQ ID NO:1239), CUUCAAGAGCAACAGUGCUG (SEQ ID NO:1240), AGAGCAACAGUGCUGUGGCC (SEQ ID NO:1241), AAAGUCAGAUUUGUUGCUCC (SEQ ID NO:1242), ACAAAACUGUGCUAGACAUG (SEQ ID NO:1243), AAACUGUGCUAGACAUG (SEQ ID NO:1244), UGUGCUAGACAUGAGGUCUA (SEQ ID NO:1245), GGCUGGGGAAGAAGGUGUCUUC (SEQ ID NO:1246), GCUGGGGAAGAAGGUGUCUUC (SEQ ID NO:1247), GGGGAAGAAGGUGUCUUC (SEQ ID NO:1248), GUUUUGUCUGUGAUAUACACAU (SEQ ID NO:1249), GGCAGACAGACUUGUCACUGGAUU (SEQ ID NO:1250), GCAGACAGACUUGUCACUGGAUU (SEQ ID NO:1251), GACAGACUUGUCACUGGAUU (SEQ ID NO:1252), GUGAAUAGGCAGACAGACUUGUCA (SEQ ID NO:1253), GAAUAGGCAGACAGACUUGUCA (SEQ ID NO:1254), GAGUCUCUCAGCUGGUACACGG (SEQ ID NO:1255), GUCUCUCAGCUGGUACACGG (SEQ ID NO:1256), GGUACACGGCAGGGUCAGGGUU (SEQ ID NO:1257), и GUACACGGCAGGGUCAGGGUU (SEQ ID NO:1258).
44. Сконструированная клетка по п.41, где гРНК содержит нацеливающий домен, который комплементарен домену-мишени гена TRBC, необязательно в одном или обоих генах TRBC1 и TRBC2, причем указанный нацеливающий домен-мишень содержит последовательность, выбранную из CACCCAGAUCGUCAGCGCCG (SEQ ID NO:1259), CAAACACAGCGACCUCGGGU (SEQ ID NO:1260), UGACGAGUGGACCCAGGAUA (SEQ ID NO:1261), GGCUCUCGGAGAAUGACGAG (SEQ ID NO:1262), GGCCUCGGCGCUGACGAUCU (SEQ ID NO:1053), GAAAAACGUGUUCCCACCCG (SEQ ID NO:1263), AUGACGAGUGGACCCAGGAU (SEQ ID NO:1264), AGUCCAGUUCUACGGGCUCU (SEQ ID NO:1265), CGCUGUCAAGUCCAGUUCUA (SEQ ID NO:1266), AUCGUCAGCGCCGAGGCCUG (SEQ ID NO:1267), UCAAACACAGCGACCUCGGG (SEQ ID NO:1268), CGUAGAACUGGACUUGACAG (SEQ ID NO:1269), AGGCCUCGGCGCUGACGAUC (SEQ ID NO:1270), UGACAGCGGAAGUGGUUGCG (SEQ ID NO:1271), UUGACAGCGGAAGUGGUUGC (SEQ ID NO:1272), UCUCCGAGAGCCCGUAGAAC (SEQ ID NO:1273), CGGGUGGGAACACGUUUUUC (SEQ ID NO:1274), GACAGGUUUGGCCCUAUCCU (SEQ ID NO:1275), GAUCGUCAGCGCCGAGGCCU (SEQ ID NO:1276), GGCUCAAACACAGCGACCUC (SEQ ID NO:1277), UGAGGGUCUCGGCCACCUUC (SEQ ID NO:1278), AGGCUUCUACCCCGACCACG (SEQ ID NO:1279), CCGACCACGUGGAGCUGAGC (SEQ ID NO:1280), UGACAGGUUUGGCCCUAUCC (SEQ ID NO:1281), CUUGACAGCGGAAGUGGUUG (SEQ ID NO:1282), AGAUCGUCAGCGCCGAGGCC (SEQ ID NO:1283), GCGCUGACGAUCUGGGUGAC (SEQ ID NO:1284), UGAGGGCGGGCUGCUCCUUG (SEQ ID NO:1285), GUUGCGGGGGUUCUGCCAGA (SEQ ID NO:1286), AGCUCAGCUCCACGUGGUCG (SEQ ID NO:1287), GCGGCUGCUCAGGCAGUAUC (SEQ ID NO:1288), GCGGGGGUUCUGCCAGAAGG (SEQ ID NO:1289), UGGCUCAAACACAGCGACCU (SEQ ID NO:1290), ACUGGACUUGACAGCGGAAG (SEQ ID NO:1291), GACAGCGGAAGUGGUUGCGG (SEQ ID NO:1292), GCUGUCAAGUCCAGUUCUAC (SEQ ID NO:1293), GUAUCUGGAGUCAUUGAGGG (SEQ ID NO:1294), CUCGGCGCUGACGAUCU (SEQ ID NO:1295), CCUCGGCGCUGACGAUC (SEQ ID NO:1296), CCGAGAGCCCGUAGAAC (SEQ ID NO:1297), CCAGAUCGUCAGCGCCG (SEQ ID NO:1298), GAAUGACGAGUGGACCC (SEQ ID NO:1299), GGGUGACAGGUUUGGCCCUAUC (SEQ ID NO:1300), GGUGACAGGUUUGGCCCUAUC (SEQ ID NO:1301), GUGACAGGUUUGGCCCUAUC (SEQ ID NO:1302), GACAGGUUUGGCCCUAUC (SEQ ID NO:1303), GAUACUGCCUGAGCAGCCGCCU (SEQ ID NO:1304), GACCACGUGGAGCUGAGCUGGUGG (SEQ ID NO:1305), GUGGAGCUGAGCUGGUGG (SEQ ID NO:1306), GGGCGGGCUGCUCCUUGAGGGGCU (SEQ ID NO:1307), GGCGGGCUGCUCCUUGAGGGGCU (SEQ ID NO:1308), GCGGGCUGCUCCUUGAGGGGCU (SEQ ID NO:1309), GGGCUGCUCCUUGAGGGGCU (SEQ ID NO:1310), GGCUGCUCCUUGAGGGGCU (SEQ ID NO:1311), GCUGCUCCUUGAGGGGCU (SEQ ID NO:1312), GGUGAAUGGGAAGGAGGUGCACAG (SEQ ID NO:1313), GUGAAUGGGAAGGAGGUGCACAG (SEQ ID NO:1314), и GAAUGGGAAGGAGGUGCACAG (SEQ ID NO:1315).
45. Сконструированная клетка по п.44, отличающаяся тем, что:
сконструированная клетка содержит генетическое нарушение гена константной области Т-клеточного рецептора альфа (TRAC) и гена константной области Т-клеточного рецептора бета (TRBC); и/или
один или более агентов содержат агент, содержащий, по меньшей мере, одну гРНК, имеющую нацеливающий домен, который комплементарен домену-мишени гена TRAC, и агент, содержащий, по меньшей мере, одну гРНК, имеющую нацеливающий домен, который комплементарен домену-мишени гена TRBC, необязательно одного или обоих, гена TRBC1 и гена TRBC2.
46. Сконструированная клетка по п.40, где нацеливающий домен содержит последовательность, комплементарную домену-мишени гена TRAC, и нацеливающий домен содержит последовательность GAGAAUCAAAAUCGGUGAAU (SEQ ID NO: 1048).
47. Сконструированная клетка по п.40, где нацеливающий домен содержит последовательность, комплементарную домену-мишени гена TRBC, и нацеливающий домен содержит последовательность GGCCUCGGCGCUGACGAUCU (SEQ ID NO: 1053).
48. Сконструированная клетка по п.40, где гРНК дополнительно содержит первый домен комплементарности, второй домен комплементарности, который комплементарен первому домену комплементарности, проксимальный домен и, необязательно, хвостовой домен.
49. Сконструированная клетка по п.48, где первый домен комплементарности и второй домен комплементарности соединены связывающим доменом.
50. Сконструированная клетка по п.48 или 49, где гид-РНК содержит 3'-поли-А-хвост и 5'-анти-обратный Cap-аналог.
51. Сконструированная клетка по п.42, где молекула Cas9 является ферментативно активной Cas9.
52. Сконструированная клетка по п.42, где молекула Cas9 представляет собой молекулу Cas9 S. aureus.
53. Сконструированная клетка по п.42, где молекула Cas9 представляет собой Cas9 S. pyogenes.
54. Сконструированная клетка по п.42, где сконструированная клетка содержит генетическое нарушение локуса константной области Т-клеточного рецептора альфа (TRAC).
55. Сконструированная клетка по п.42, где эндогенный локус TRAC дополнительно модифицируется путем интеграции последовательности нуклеиновой кислоты, кодирующей TCR или его антигенсвязывающий фрагмент, в локус TRAC, необязательно через HDR.
56. Сконструированная клетка по п.42, где эндогенный локус TRAC дополнительно модифицируется путем интеграции трансгенной последовательности, кодирующей часть TCR или его антигенсвязывающего фрагмента, необязательно посредством направляемой гомологией репарации (HDR).
57. Сконструированная клетка по любому из пп.55 или 56, где TCR или его антигенсвязывающий фрагмент содержит Cα, причем по меньшей мере, часть указанной Cα кодируется открытой рамкой считывания или ее частичной последовательностью эндогенного локуса TRAC.
58. Сконструированная клетка по п.55, где модифицированный локус TRAC содержит слияние в рамке (i) трансгенной последовательности, кодирующей часть TCR, и (ii) открытой рамки считывания или ее частичной последовательности эндогенного локуса TRAC.
59. Сконструированная клетка по п.56 или 58, где трансгенная последовательность не содержит последовательности, кодирующей 3'-UTR или интрон.
60. Сконструированная клетка по п.57 или 58, где открытая рамка считывания или ее частичная последовательность содержит 3'-UTR эндогенного локуса TRAC.
61. Сконструированная клетка по п.56, где трансгенная последовательность интегрирована в 3'-области относительно крайнего 5'-нуклеотида экзона 1 и в 5'-области относительно крайнего 3'-нуклеотида экзона 1 открытой рамки считывания эндогенного локуса TRAC.
62. Сконструированная клетка по п.57, где по меньшей мере часть Cα кодируется по меньшей мере экзонами 2-4 открытой рамки считывания эндогенного локуса TRAC.
63. Сконструированная клетка по п.57, отличающаяся тем, что, по меньшей мере, часть Cα кодируется, по меньшей мере, частью экзона 1 и экзонов 2-4 открытой рамки считывания эндогенного локуса TRAC.
64. Сконструированная клетка по п.56, где трансгенная последовательность кодирует бета-цепь Т-клеточного рецептора (TCRβ) и/или вариабельную область TCR альфа (Vα).
65. Сконструированная клетка по п.54, дополнительно содержащая генетическое нарушение локуса константной области Т-клеточного рецептора бета (TRBC).
66. Сконструированная клетка по п.56, где трансгенная последовательность или последовательность нуклеиновой кислоты, кодирующая TCR или его антигенсвязывающий фрагмент, содержит один или более полицистронных элементов.
67. Сконструированная клетка по п.66, где один или более полицистронных элементов находятся в 5'-области от трансгенной последовательности или последовательности нуклеиновой кислоты, кодирующей TCR или его антигенсвязывающий фрагмент.
68. Сконструированная клетка по п.66, где полистронный элемент(элементы) расположен между последовательностью нуклеиновой кислоты, кодирующей TCRα или его часть, и последовательностью нуклеиновой кислоты, кодирующей TCRβ или его часть.
69. Сконструированная клетка по п.66, где один или более полицистронных элементов представляют собой или содержат последовательность пропуска рибосомы, где последовательность пропуска рибосомы представляет собой элемент T2A, P2A, E2A или F2A.
70. Сконструированная клетка по п.56, где трансгенная последовательность или последовательность нуклеиновой кислоты, кодирующая TCR или его антигенсвязывающий фрагмент, содержит один или более гетерологичных или регуляторных контрольных элементов, функционально связанных с контролем экспрессии TCR, когда он экспрессируется сконструированной клеткой.
71. Сконструированная клетка по п.70, где один или более гетерологичных регуляторных или контрольных элементов содержат промотор, энхансер, интрон, сигнал полиаденилирования, консенсусную последовательность Козака, акцепторную последовательность сплайсинга и/или донорную последовательность сплайсинга.
72. Сконструированная клетка по п.70 или 71, где гетерологичный регуляторный или контрольный элемент содержит гетерологичный промотор, необязательно промотор фактора элонгации 1 альфа (EF1α) человека или промотор MND или его вариант.
73. Сконструированная клетка по п.35, где TCR или его антигенсвязывающий фрагмент или его часть являются гетерологичными по отношению к клетке.
74. Сконструированная клетка по п.35, где сконструированная клетка представляет собой первичную клетку, полученную от пациента.
75. Сконструированная клетка по п.74, где пациентом является млекопитающее.
76. Сконструированная клетка по п.74 или 75, где пациентом является человек.
77. Сконструированная клетка по п.35, где Т-клетка представляет собой CD8+ Т-клетку.
78. Сконструированная клетка по п.35, где Т-клетка представляет собой CD4+ Т-клетку.
79. Способ получения клетки по любому из пп.35-78, включающий введение вектора по любому из пп.26-34 в клетку in vitro или ex vivo.
80. Способ получения клетки, включающий введение полинуклеотида по любому из пп.1-25 или вектора по любому из пп.26-34 в клетку in vitro или ex vivo, где указанная клетка представляет собой Т-клетку.
81. Способ по п.79 или 80, где вектор представляет собой вирусный вектор, а введение осуществляют трансдукцией.
82. Способ по п.81, дополнительно включающий введение в клетку одного или более агентов, где каждый из одного или более агентов независимо способен индуцировать генетическое нарушение гена константной области Т-клеточного рецептора альфа (TRAC) и/или гена константной области Т-клеточного рецептора бета (TRBC).
83. Способ получения сконструированной Т-клетки, включающий:
(i) введение полинуклеотида по п.1 или вектора по п.26 в клетку; и
(ii) введение в клетку одного или более агентов, где каждый из одного или более агентов независимо способен индуцировать генетическое нарушение гена константной области Т-клеточного рецептора альфа (TRAC) и/или гена константной области Т-клеточного рецептора бета (TRBC).
84. Способ по п.83, где один или более агентов, способных индуцировать генетическое нарушение, включают ДНК-связывающий белок или ДНК-связывающую нуклеиновую кислоту, которые специфически связываются с сайтом-мишенью или гибридизуются с ним.
85. Способ по п.84, где один или более агентов, способных индуцировать генетическое нарушение, включают (a) слитый белок, содержащий ДНК-нацеленный белок и нуклеазу, или (b) РНК-направляемую нуклеазу.
86. Способ по п.85, где ДНК-нацеленный белок или РНК-направляемая нуклеаза содержат белок цинкового пальца (ZFP), белок TAL или нуклеазу Cas, ассоциированную с нуклеиновой кислотой CRISPR (короткие палиндромные повторы, регулярно расположенные группами), специфичную для сайта-мишени в гене TRAC и/или TRBC.
87. Способ по п.86, где один или более агентов включают нуклеазу цинкового пальца (ZFN), эффекторную нуклеазу TAL (TALEN) или комбинацию CRISPR-Cas9, которые специфически связываются, распознают или гибридизуются с сайтом-мишенью.
88. Способ по п.86 или 87, где каждый из одного или более агентов содержит гид-РНК (гРНК), имеющую нацеливающий домен, который комплементарен по меньшей мере одному сайту-мишени.
89. Способ по п.83, где на генетическое нарушение воздействует один или более агентов, которые включают (а) по меньшей мере одну гРНК, имеющую нацеливающий домен, который комплементарен домену-мишени гена TRAC и/или TRBC, или (b) по меньшей мере одну нуклеиновую кислоту, кодирующую по меньшей мере одну гРНК.
90. Способ по п.89, где один или более агентов вводят в виде рибонуклеопротеинового комплекса (RNP), содержащего гРНК и белок Cas9.
91. Способ по п.90, где RNP вводят посредством электропорации, генной пушки, трансфекции фосфатом кальция, сжатия или сдавливания клеток.
92. Способ по п.90 или 91, где RNP вводят посредством электропорации.
93. Способ по п.88, где один или более агентов вводят в виде одного или более полинуклеотидов, кодирующих гРНК и/или белок Cas9.
94. Способ по п.82, где один или более агентов содержат по меньшей мере один комплекс молекулы Cas9 и по меньшей мере одну гРНК, имеющую нацеливающий домен, который комплементарен домену-мишени гена TRAC и/или гена TRBC.
95. Способ по п.82, где один или более агентов содержат, по меньшей мере, один комплекс из молекулы Cas9 и гРНК, имеющей нацеливающий домен, который представляет собой один или оба из (1) комплементарного домену-мишени гена TRAC, причем указанный нацеливающий домен содержит последовательность, выбранную из любой из SEQ ID NO: 1048 и 1229-1258; и (2) комплементарного домену-мишени гена TRBC, необязательно одного или обоих из генов TRBC1 и TRBC2, причем указанный нацеливающий домен содержит последовательность, выбранную из любой из SEQ ID NO: 1053 и 1259-1315.
96. Способ по п.89, где нацеливающий домен содержит последовательность, комплементарную домену-мишени гена TRAC, и нацеливающий домен содержит последовательность GAGAAUCAAAAUCGGUGAAU (SEQ ID NO: 1048).
97. Способ по п.89, где нацеливающий домен содержит последовательность, комплементарную домену-мишени гена TRBC, и нацеливающий домен содержит последовательность GGCCUCGGCGCUGACGAUCU (SEQ ID NO: 1053).
98. Способ по п.89, где гид-РНК дополнительно содержит первый домен комплементарности, второй домен комплементарности, который комплементарен первому домену комплементарности, проксимальный домен и, необязательно, хвостовой домен.
99. Способ по п.98, где первый домен комплементарности и второй домен комплементарности соединены связывающим доменом.
100. Способ по п.99, где гид-РНК содержит 3'-поли-А-хвост и 5'- анти-обратный Cap-аналог.
101. Способ по п.96, где молекула Cas9 представляет собой ферментативно активную Cas9.
102. Способ по п.96, где молекула Cas9 представляет собой молекулу Cas9 S. aureus.
103. Способ по п.96, где молекула Cas9 представляет собой Cas9 S. pyogenes.
104. Способ по п.82, отличающийся тем, что, по меньшей мере или более чем 35%, 40%, 45%, 50%, 55%, 60%, 65%, 70%, 75%, 80%, 90% или 95% клеток во множестве сконструированных клеток содержат генетическое нарушение в гене TRAC и/или гене TRBC.
105. Способ по п.82, где по меньшей мере или более чем 90%, 95%, 96%, 97% или 98% клеток во множестве сконструированных клеток содержат генетическое нарушение в гене TRAC и/или гене TRBC.
106. Способ по п.79, где по меньшей мере или более чем 5%, 10%, 15%, 20%, 25%, 30%, 40%, 50% или более клеток во множестве сконструированных клеток экспрессируют введенный TCR или его антигенсвязывающий фрагмент и/или проявляют антигенное связывание с белком HPV, необязательно HPV E6 или HPV E7.
107. Способ по п.79, где по меньшей мере или более чем 5%, 10%, 15%, 20%, 25%, 30%, 40%, 50% или более клеток во множестве сконструированных клеток экспрессируют введенный TCR или его антигенсвязывающий фрагмент и/или проявляют антигенное связывание с белком HPV, необязательно HPV E6 или HPV E7.
108. Способ по п.79, где по меньшей мере или более чем 5%, 10%, 15%, 20%, 25%, 30%, 40%, 50% или более клеток во множестве сконструированных клеток экспрессируют введенный TCR или его антигенсвязывающий фрагмент и/или проявляют антигенное связывание с белком HPV, необязательно HPV E6 или HPV E7.
109. Способ по п.82, где один или более агентов и молекулу нуклеиновой кислоты, полинуклеотид или вектор вводят одновременно или последовательно в любом порядке.
110. Способ по п.82, где молекулу нуклеиновой кислоты, полинуклеотид или вектор вводят после введения одного или более агентов.
111. Способ по п.110, где молекулу нуклеиновой кислоты, полинуклеотид или вектор вводят сразу после или в течение примерно 30 секунд, 1 минуты, 2 минут, 3 минут, 4 минут, 5 минут, 6 минут, 6 минут, 8 минут, 9 минут, 10 минут, 15 минут, 20 минут, 30 минут, 40 минут, 50 минут, 60 минут, 90 минут, 2 часов, 3 часов или 4 часов после введения агента.
112. Фармацевтическая композиция для лечения заболевания или расстройства, ассоциированных с HPV16, содержащая сконструированную клетку по любому из пп.35-78 и фармацевтически приемлемое вспомогательное вещество.
113. Фармацевтическая композиция для лечения заболевания или расстройства, ассоциированных с HPV16, содержащая сконструированную клетку, полученную путем трансформации полинуклеотидом по любому из пп.1-25 и фармацевтически приемлемое вспомогательное вещество.
114. Композиция по п.112 или 113, отличающаяся тем, что:
по меньшей мере 70%, 75%, 80%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% сконструированных клеток в композиции содержат генетическое разрушение эндогенного гена константной области Т-клеточного рецептора альфа (TRAC) и/или гена константной области Т-клеточного рецептора бета (TRBC); и/или
по меньшей мере 80%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% сконструированных клеток в композиции не экспрессируют или не экспрессируют на детектируемом уровне генный продукт эндогенного гена TRAC или TRBC.
115. Композиция по п.112 или 113, где по меньшей мере или более чем 90%, 95%, 96%, 97% или 98% клеток в композиции содержат генетическое нарушение эндогенного гена TRAC и/или гена TRBC и/или не экспрессируют или не экспрессируют на детектируемом уровне генный продукт эндогенного гена TRAC или TRBC.
116. Композиция по п.112 или 113, где по меньшей мере или более чем 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97% или 98% клеток в композиции i) экспрессируют сконструированный или рекомбинантный TCR или его антигенсвязывающий фрагмент и (ii) содержат генетическое нарушение эндогенного гена TRAC и/или гена TRBC и/или не экспрессируют или не экспрессируют на детектируемом уровне генный продукт эндогенного гена TRAC или TRBC.
117. Композиция по п.114, где ген TRBC представляет собой один или оба из гена константной области 1 Т-клеточного рецептора бета (TRBC1) или гена константной области 2 Т-клеточного рецептора бета (TRBC2).
118. Композиция по п.114, где генный продукт представляет собой мРНК или белок, кодируемый геном TRAC или TRBC.
119. Композиция по п.112, где сконструированные клетки содержат CD4+ и/или CD8+ Т-клетки.
120. Композиция по п.112, где сконструированные клетки содержат CD4+ и CD8+ Т-клетки.
121. Фармацевтическая композиция для лечения заболевания или расстройства, ассоциированных с HPV16, содержащая сконструированную CD8+ клетку по п.77 и сконструированную CD4+ клетку по п.78 и фармацевтически приемлемое вспомогательное вещество.
122. Композиция по п.112, где TCR или его антигенсвязывающий фрагмент связываются или распознают пептидный эпитоп HPV 16 в контексте молекулы МНС, что является по меньшей мере частично CD8-независимым.
123. Композиция по п.119, где CD8+ клетки и CD4+ клетки сконструированы с одним и тем же TCR или его антигенсвязывающим фрагментом, и/или каждая из них сконструирована с TCR или его антигенсвязывающим фрагментом, которые связываются или распознают тот же пептидный эпитоп HPV 16 в контексте молекулы МНС.
124. Композиция по п.114, отличающаяся тем, что:
генетическое нарушение включает мутацию или делецию в области гена TRAC или TRBC, которая находится в кодирующей области, необязательно в ранней кодирующей области гена, находится в пределах экзона 1 гена, находится в кодирующей области в пределах 500, 400 300, 200, 100 или 50 пар оснований от старт-кодона гена, находится в последовательности сайта-мишени, которая комплементарна нацеливающему сайту нацеливающего домена гРНК, имеющего последовательность, выбранную из любой из SEQ ID NO: 1053 и 1259-1315, и/или с которым специфически гибридизуется нацеливающий домен, имеющий последовательность, выбранную из SEQ ID NO: 1053 и 1259-1315, и/или находится в последовательности сайта-мишени, которая комплементарна нацеливающему сайту нацеливающего домена гРНК, имеющего последовательность, выбранную из любой из SEQ ID NO: 1048 и 1229-1258, и/или с которым специфически гибридизуется нацеливающий домен, имеющий последовательность, выбранную из SEQ ID NO: 1048 и 1229-1258.
125. Композиция по п.121, где по меньшей мере одна из сконструированных клеток представляет собой клетку по любому из пп.35-80.
126. Композиция по п.112, где на генетическое нарушение воздействует один или более агентов, которые содержат (a) по меньшей мере одну гРНК, имеющую нацеливающий домен, который комплементарен домену-мишени гена TRAC и/или гена TRBC, или (b) по меньшей мере одну нуклеиновую кислоту, кодирующую по меньшей мере одну гРНК.
127. Композиция по п.126, где один или более агентов вводят в виде рибонуклеопротеинового (RNP) комплекса, содержащего гРНК и белок Cas9.
128. Композиция по п.127, где RNP вводят посредством электропорации, генной пушки, трансфекции фосфатом кальция, сжатия или сдавливания клеток.
129. Композиция по п.127 или п.128, где RNP вводят электропорацией.
130. Композиция по п.126, где один или более агентов вводят в виде одного или более полинуклеотидов, кодирующих гРНК и/или белок Cas9.
131. Композиция по п.126, где один или более агентов содержат по меньшей мере один комплекс из молекулы Cas9 и по меньшей мере одной гРНК, имеющей нацеливающий домен, который комплементарен домену-мишени гена TRAC и/или гена TRBC.
132. Композиция по п.126, где один или более агентов содержат по меньшей мере один комплекс из молекулы Cas9 и гРНК, имеющей нацеливающий домен, который представляет собой один или оба из следующего: (1) комплементарен домену-мишени гена TRAC, причем указанный нацеливающий домен содержит последовательность, выбранную из любой из SEQ ID NO: 1048 и 1229-1258; и (2) комплементарен домену-мишени гена TRBC, необязательно одного или обоих из генов TRBC1 и TRBC2, причем указанный нацеливающий домен содержит последовательность, выбранную из любой из SEQ ID NO: 1053 и 1259-1315.
133. Композиция по п.126, где нацеливающий домен содержит последовательность, комплементарную домену-мишени гена TRAC, и нацеливающий домен содержит последовательность GAGAAUCAAAAUCGGUGAAU (SEQ ID NO: 1048).
134. Композиция по п.126, где нацеливающий домен содержит последовательность, комплементарную домену-мишени гена TRBC, и нацеливающий домен содержит последовательность GGCCUCGGCGCUGACGAUCU (SEQ ID NO: 1053).
135. Композиция по п.126, где гид-РНК дополнительно содержит первый домен комплементарности, второй домен комплементарности, который комплементарен первому домену комплементарности, проксимальный домен и, необязательно, хвостовой домен.
136. Композиция по п.135, где первый домен комплементарности и второй домен комплементарности соединены с помощью связывающего домена.
137. Композиция по п.136, где гид-РНК содержит 3'-поли-А-хвост и 5'-анти-обратный Cap-аналог.
138. Композиция по п.126, где молекула Cas9 представляет собой ферментативно активную Cas9.
139. Композиция по п.126, где молекула Cas9 представляет собой молекулу Cas9 S. aureus.
140. Композиция по п.126, где молекула Cas9 представляет собой Cas9 S. pyogenes.
141. Способ лечения заболевания или расстройства, ассоциированных с HPV16, у пациента, включающий введение пациенту сконструированной клетки по п.35.
142. Способ лечения заболевания или расстройства, ассоциированных с HPV16, у пациента, включающий введение пациенту композиции по п.112.
143. Способ по п.141, где заболевание или расстройство представляет собой онкологическое заболевание.
144. Способ по п.141, где пациентом является человек.
145. Композиция по п.112 для применения при лечении заболевания или расстройства, ассоциированных с HPV16, у субъекта.
146. Применение композиции по п.112 для изготовления лекарственного средства для лечения заболевания или расстройства, ассоциированных с HPV16, у субъекта.
147. Композиция по п.145 или применение по п.146, где заболевание или расстройство представляют собой онкологическое заболевание.
148. Композиция по п.145 или применение по п.146, где субъект является человеком.
| WO2015184228 A1, 03.12.2015 | |||
| WO2016146618 A1, 22.09.2016 | |||
| LINDSEY M | |||
| DRAPER et al., Targeting of HPV-16+ epithelial cancer cells by TCR gene engineered T cells directed against E6, Clin Cancer Res, 2015, Vol.21, N.19, pp | |||
| Устройство для контролирования состояния заказов в производстве | 1926 |
|
SU4431A1 |
| САЛЯЕВ Р.К.и др., Исследование иммуногенности антигенного белка высокоонкогенного вируса папилломы человека HPV16 | |||
Авторы
Даты
2023-10-03—Публикация
2018-09-28—Подача