ОБЛАСТЬ ТЕХНИКИ
Настоящее изобретение направлено на обеспечение конструкций нуклеиновых кислот и белков, которые вовлечены или воздействуют на метаболические пути, опосредующие или влияющие на метаболизм клетки, например, на перемещение через мембрану эндоплазматического ретикулума (ЭР) и/или секрецию через цитоплазматическую мембрану, а также на способы влияния на клеточный метаболизм. Настоящее изобретение также направлено на получение и применение рекомбинантных клеток млекопитающих, в которых, например, модифицированы перемещение/секреция большого разнообразия гетерологичных белков (продуктов экспрессии трансгенов). Указанные способы, конструкции нуклеиновых кислот, как правило, разработаны таким образом, чтобы улучшить экспрессию трансгена.
УРОВЕНЬ ТЕХНИКИ
Получение терапевтических белков биотехнологическими способами, а также генная и клеточная терапия зависят от успешной экспрессии трансгенов, внедренных в эукариотическую клетку. Для успешной экспрессии трансгена, как правило, требуется встраивание трансгена в хромосому хозяина, при этом указанная экспрессия ограничена, среди прочего, количеством встроенных копий трансгена и эпигенетическими эффектами, которые могут вызывать низкий уровень или нестабильность транскрипции и/или высокую клональную изменчивость. Недостаточный или пониженный транспорт продукта экспрессии трансгена из клетки также часто ограничивает получение терапевтических белков, а также генную и клеточную терапию.
Публикации и другие материалы, включая патенты, заявки на патент и номера доступа, используемые в данной заявке для иллюстрации настоящего изобретения и, в частности, для предоставления дополнительных подробностей касательно осуществления настоящего изобретения, полностью включены в данную заявку посредством ссылки.
Для повышения и стабилизации экспрессии трансгена в клетках млекопитающих все чаще применяют эпигенетические регуляторы, для того чтобы защитить трансгены от отрицательных эффектов положения (Bell и Felsenfeld, 1999). Данные эпигенетические регуляторы включают граничные или инсуляторные элементы, регуляторные области локусов (LCR), стабилизирующие и антирепрессорные (STAR) элементы, универсальные элементы, открывающие хроматин (UCOE), и упомянутые выше участки прикрепления к матриксу (MAR). Все эти эпигенетические регуляторы применялись для получения рекомбинантного белка в линиях клеток млекопитающих (Zahn-Zabal и др., 2001; Kim и др., 2004) и в способах генной терапии (Agarwal и др., 1998; Allen и др., 1996; Castilla и др., 1998).
Процессинг и транспорт из клетки продукта экспрессии трансгена часто сопряжен с различными затруднениями. Клетка, которая снабжена лишь средствами для процессинга и транспорта ее нативных белков, может быстро испытать перегрузку от транспорта некоторых типов продуктов экспрессии трансгенов, особенно когда они продуцируются на аномально высоких уровнях, что часто желательно, что приводит к агрегации продукта внутри клетки и/или, например, препятствует правильному фолдингу функционального белкового продукта.
Для преодоления затруднений, связанных с транспортом и процессингом, использовались различные подходы. Например, сконструированы клетки СНО с улучшенными секреторными свойствами путем экспрессии белков семейства SM Munc18c или Sly1, которые действуют как регуляторы транспорта мембранных везикул и, следовательно, экзоцитоза секретируемых белков (публикация заявки на патент США 20090247609). Х-бокс-связывающий белок 1 (Xbp1) - фактор транскрипции, который регулирует дифференцировку секреторных клеток и поддержание и экспансию ЭР, или различные протеиндисульфидизомеразы (PDI) применяли для снижения стресса ЭР и повышения секреции белка (Mohan и др. 2007). Другие попытки повышения секреции белка включали экспрессию шаперонов ERp57, кальнексина, кальретикулина и BiP1 в клетках СНО (Chung и др., 2004). Было показано, что экспрессия белка, индуцированного холодовым шоком, в частности, индуцированного холодом РНК-связывающего белка (CIRP), повышает выход рекомбинантного гамма-интерферона. Также были предприняты попытки сверхэкспрессировать белки секреторных комплексов. Тем не менее, например, Lakkaraju и др. (2008) сообщили, что экспрессия экзогенного SRP14 в человеческих клетках дикого типа (например, в клетках, которые не были сконструированы для экспрессии низких уровней SRP14) не улучшала эффективность секреции секретируемого белка щелочной фосфатазы.
Таким образом, существует потребность в эффективной, надежной экспрессии трансгена, например, при получении рекомбинантного белка и для генной терапии. Также существует потребность в успешном транспорте продукта экспрессии трансгена за пределы клетки.
На удовлетворение данной и других потребностей, существующих в данной области техники, направлены варианты реализации настоящего изобретения.
КРАТКОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
Настоящее изобретение в одном варианте реализации направлено на рекомбинантную молекулу нуклеиновой кислоты, содержащую:
(a) 5'- и 3'-транспозон-специфичный инвертированный концевой повтор (ITR),
(b) по меньшей мере одну последовательность нуклеиновой кислоты, кодирующую белок экспрессии/процессинга трансгена (ТЕР), или функциональную РНК ТЕР, расположенную между 5'- и 3'-ITR, которая находится под контролем промотора, и
(c) возможно по меньшей мере один трансген, также расположенный между 5'- и 3'-ITR, который находится под контролем трансгенного промотора, при этом указанная молекула нуклеиновой кислоты необязательно является частью вектора.
Указанная рекомбинантная молекула нуклеиновой кислоты может содержать по меньшей мере один эпигенетический регуляторный элемент, в частности, по меньшей мере один элемент MAR (участок прикрепления к матриксу).
Элемент MAR может быть расположен между 5'- и 3'-ITR. Трансген, такой как ген устойчивости к антибиотику или ген, кодирующий иммуноглобулин, возможно под контролем дополнительного промотора, может быть расположен между 5'- и 3'-ITR, например, между 5'-ITR и MAR.
Белок ТЕР или функциональная РНК ТЕР может представлять собой белок или функциональную РНК, которая непосредственно или опосредованно вовлечена во встраивание последовательностей нуклеиновых кислот в геном, в процессинг или трансляцию продукта трансгенной РНК или участвует в перемещении в ЭР, секреции, процессинге, фолдинге, транспорте ЭР-Гольджи-плазматическая мембрана, гликозилировании и/или другой посттрансляционной модификации белков, таких как продукты экспрессии трансгенов.
Белок ТЕР может представлять собой белок пути секреции белка, белок путей рекомбинации или репарации ДНК, белок процессинга белка или метаболический белок, включая шапероны, такие как BiP, или комбинацию перечисленных белков.
Белок ТЕР может представлять собой один или более из следующих белков пути секреции белка: SRP14 человека (hSRP14), hSec61α1, hSec61β, hSec61γ, hSRP54, hSRP9, hSRPRα, hSRPβ и hCANX.
Белок ТЕР также может соответствовать одной или более из следующих последовательностей аминокислот белков пути секреции белка: hSRP14 с последовательностью SEQ ID NO: 13, hSec61α1 с последовательностью SEQ ID NO: 15, hSec61β с последовательностью SEQ ID NO: 17, hSec61γ с последовательностью SEQ ID NO: 19, hSRP54 с последовательностью SEQ ID NO: 21, hSRP9 с последовательностью SEQ ID NO: 23, hSRPRα с последовательностью SEQ ID NO: 25, hSRPβ с последовательностью SEQ ID NO: 27 и hCANX с последовательностью SEQ ID NO: 29 и/или может соответствовать последовательностям аминокислот, идентичным более чем на 80%, 90%, 95% или 98% указанным последовательностям.
Белок ТЕР может представлять собой один или более из следующих белков процессинга белка или метаболических белков: hUCP4, hCMPSAT, ST6Gal1 крысы (rST6Gal1), hCOMSC, Т-синтазу человека, hP4HA1, hP4HB, hGILZ, hCyPB, hNRF2, hHK1, hPDI, hPIN1, hSEPW1, hCALR, hDDOST, hHSP40, hATP5A1, hSERCA2, hPDIA4, hHSC70/HSPA8, hHYOU1, hCMP-SAS, беклин-1 человека, hERdj3, CHO-AGE, hWipl, hRTP4, hREEP2, hDPM1 и hDRiP78.
Белок ТЕР также может соответствовать одной или более из следующих последовательностей аминокислот белков процессинга белка или метаболических белков: hUCP4 с последовательностью SEQ ID NO: 31, hCMPSAT с последовательностью SEQ ID NO: 33, rST6GaI1 с последовательностью SEQ ID NO: 35, hCOMSC с последовательностью SEQ ID NO: 37, Т-синтазы человека с последовательностью SEQ ID NO: 39, hP4HA1 с последовательностью SEQ ID NO: 41, hP4HB с последовательностью SEQ ID NO: 43, hGILZ с последовательностью SEQ ID NO: 45, hCyPB с последовательностью SEQ ID NO: 47, hNRF2 с последовательностью SEQ ID NO: 49, hHK1 с последовательностью SEQ ID NO: 51, hPDI с последовательностью SEQ ID NO: 53, hPIN1 с последовательностью SEQ ID NO: 55, hSEPW1 с последовательностью SEQ ID NO: 57, hCALR с последовательностью SEQ ID NO: 59, hDDOST с последовательностью SEQ ID NO: 62, hHSP40 с последовательностью SEQ ID NO: 64, hATP5A1 с последовательностью SEQ ID NO: 66, hSERCA2 с последовательностью SEQ ID NO: 68, hPDIA4 с последовательностью SEQ ID NO: 70, hHSC70 /HSPA8 с последовательностью SEQ ID NO: 72, hHYOU1 с последовательностью SEQ ID NO: 74, hCMP-SAS с последовательностью SEQ ID NO: 76, беклина-1 человека с последовательностью SEQ ID NO: 78, hERdj3 с последовательностью SEQ ID NO: 80, CHO-AGE с последовательностью SEQ ID NO: 82, hWip1 с последовательностью SEQ ID NO: 84, hRTP4 с последовательностью SEQ ID NO: 86, hREEP2 с последовательностью SEQ ID NO: 88, hDPM1 с последовательностью SEQ ID NO: 90 и hDRiP78 с последовательностью SEQ ID NO: 92 и/или может соответствовать последовательностям аминокислот, идентичным более чем на 80%, 90%, 95% или 98% указанным последовательностям.
Белок ТЕР может представлять собой шаперон, в частности, белок BiP, а конкретнее, модифицированное производное белка BiP дрозофилы (DroBiP), последовательность которого идентична на 80%, 90%, 95% или 100% последовательности SEQ ID NO: 60.
Элемент MAR можно выбрать из последовательностей SEQ ID NO: 1 (MAR 1-68), 2 (MAR 1_6), 3 (MAR X_S29), 4 (MAR S4), 5 (MAR лизоцима цыпленка), или он предпочтительно представляет собой сконструированный, в частности, перестроенный аналог, и/или его последовательность по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентична любой из последовательностей SEQ ID NO: с 1 по 5 или любой из последовательностей SEQ ID NO: с 6 по 10.
Функциональная РНК ТЕР внутри указанной клетки может включать/состоять из последовательности нуклеиновых кислот, кодирующей функциональную РНК, предпочтительно микроРНК или короткую шпилечную РНК (кшРНК), которая препятствует экспрессии по меньшей мере одного белка пути рекомбинации или репарации ДНК, такого как, но не ограничиваясь перечисленными: Rad51, Rad51B, Rad51C, Rad51D, Xrcc2, Xrcc3, Rad52, Rad54, Brca1, Brca2, циклин D1, Ercc, MDC1, Bard1, лигаза 1, Mre11 и/или 53BP1.
Функциональная РНК ТЕР также может препятствовать экспрессии генов, последовательность которых по меньшей мере на 80%, 90%, 95%, 98% или 100% идентична Rad51 с последовательностью SEQ ID NO: 93, Rad51B с последовательностью SEQ ID NO: 94, Rad51C с последовательностью SEQ ID NO: 95, Rad51D с последовательностью SEQ ID NO: 96, Xrcc2 с последовательностью SEQ ID NO: 99, Xrcc3 с последовательностью SEQ ID NO: 100, Rad52 с последовательностью SEQ ID NO: 97, Rad54 с последовательностью SEQ ID NO: 98, Brca1 с последовательностью SEQ ID NO: 101, Brca2 с последовательностью SEQ ID NO: 102, циклину D1 с последовательностью SEQ ID NO: 103, Ercc1 с последовательностью SEQ ID NO: 104, MDC1 с последовательностью SEQ ID NO: 105, Bard1 с последовательностью SEQ ID NO: 106, лигазе 1 с последовательностью SEQ ID NO: 107, Mre11 с последовательностью SEQ ID NO: 108 и/или 53BP1 с последовательностью SEQ ID NO: 109.
Длина рекомбинантной молекулы нуклеиновой кислоты может составлять по меньшей мере 5000, 6000, 7000, 8000, 90000 или 10000 п.о.
5'- и 3'-ITR могут представлять собой 5'- и 3'-ITR транспозона Спящая Красавица или предпочтительно транспозона PiggyBac.
После первой трансфекции клетки млекопитающего одной из указанных рекомбинантных молекул нуклеиновых кислот и второй последующей трансфекции дополнительной рекомбинантной молекулой нуклеиновой кислоты, содержащей трансген, встраивание и/или экспрессия трансгена могут быть повышены в указанной клетке по сравнению с клеткой, не подвергнутой указанной первой трансфекции.
Последовательность, кодирующая ТЕР, или функциональная РНК ТЕР, упомянутые в данной заявке, могут представлять собой часть вектора, включая вектор экспрессии. Указанный вектор может содержать единичный элемент MAR, два или более элементов MAR, при этом указанный элемент(ы) может быть расположен между 5'- и 3'-ITR.
Например, указанный вектор может содержать два элемента MAR. Первый элемент MAR может быть расположен в 3'-5' направлении от ТЕР или функциональной РНК ТЕР и второй элемент MAR может быть расположен в 5'-3' направлении от ТЕР или функциональной РНК ТЕР, при этом указанный первый элемент MAR может включать элемент MAR 1_6 и/или элемент, последовательность которого по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентична последовательности SEQ ID NO: 2, в частности, перестроенные MAR на основе MAR 1-6, а конкретнее, элементы, последовательности которых по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентичны последовательности SEQ ID NO: 8 (MAR 1_6R2), и второй элемент MAR может включать элемент MAR 1-68 и/или элемент, последовательность которого по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентична последовательности SEQ ID NO: 1.
Вектор также может содержать единичный элемент MAR. Указанный единичный элемент MAR может быть расположен в 5'-3' направлении от ТЕР или функциональной РНК ТЕР, при этом указанный единичный элемент MAR может представлять собой MAR 1-68, или элемент MAR Х-29, и/или элемент, последовательность которого по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентична последовательности SEQ ID NO: 1 или 3, в частности, перестроенный MAR на основе MAR 1-68 или MAR Х-29, в частности, элемент, последовательность которого по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентична последовательности SEQ ID NO: 6, 7 или 10 (MAR 1_68R, 1_68R2 или X_29R3) или 9, и может предпочтительно представлять собой элемент MAR Х-29 и/или элемент, последовательность которого по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентична последовательности SEQ ID NO: 3.
ТЕР или функциональная РНК ТЕР может находиться под контролем промотора EF1-альфа, и за ним возможно следует поли(А) сигнал гормона роста быка (BGH).
Вектор может содержать промотор(ы) и/или эмхансер(ы) или продукты их слияния, такие как промотор(ы) и/или энхансер(ы) GAPDH, SV40p, CMV, EF1-альфа клеток СНО, Actb СНО и/или Hspa5 СНО, или сконструированные продукты их слияния, такие как CGAPDH.
Промоторы, которые являются частью вектора, могут представлять собой GAPDH с последовательностью SEQ ID NO: 111, SV40p с последовательностью SEQ ID NO: 114, CMVp с последовательностью SEQ ID NO: 113, EF1-альфа СНО с последовательностью SEQ ID NO: 112, Actb СНО с последовательностью SEQ ID NO: 115, Hspa5 СНО с последовательностью SEQ ID NO: 116, и/или продукты их слияния, такие как CGAPDH с последовательностью SEQ ID NO: 11, или их последовательности нуклеиновых кислот могут быть идентичны указанным последовательностям более чем на 80%, 90%, 95% или 98%.
Настоящее изобретение также направлено на способ экспрессии ТЕР или функциональной РНК ТЕР, включающий:
обеспечение рекомбинантной клетки млекопитающего, содержащей трансген, и вектор представляет собой вектор экспрессии, который экспрессирует ТЕР или функциональную РНК ТЕР, при этом ТЕР или функциональная РНК ТЕР, экспрессируемая с указанного вектора, возможно повышает экспрессию трансгена в указанной клетке млекопитающего на по меньшей мере 10%, по меньшей мере 20%, по меньшей мере 30%, по меньшей мере 40%, по меньшей мере 50%, по меньшей мере 60% или по меньшей мере 70%.
Указанный вектор может содержать единичный элемент MAR Х-29 и/или последовательность нуклеиновой кислоты, которая по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентична последовательности SEQ ID NO: 3, и при этом через более чем 4, 5, 6, 7, 8, 9, 10, 11, 12, 13 или 14 недель культивирования ТЕР или функциональная РНК ТЕР, экспрессируемая с указанного вектора, может повышать экспрессию интересующего гена на по меньшей мере 10%, по меньшей мере 20%, по меньшей мере 30%, по меньшей мере 40%, по меньшей мере 50%, по меньшей мере 60% или по меньшей мере 70%.
Настоящее изобретение также направлено на рекомбинантную клетку млекопитающего, содержащую не более чем 20, 15, 10 или 5 рекомбинантных молекул нуклеиновых кислот, предпочтительно встроенных в геном указанной клетки в виде единичных копий.
Выше отмечено, что белок ТЕР может представлять собой один или более из следующих белков пути секреции белка: hSRP14, hSec61α1, hSec61β, hSec61γ, hSRP54, hSRP9, hSRPRα, hSRPβ и hCANX.
Белок ТЕР также может соответствовать одной или более из следующих последовательностей аминокислот белков пути секреции белка: hSRP14 с последовательностью SEQ ID NO: 13, hSec61α1 с последовательностью SEQ ID NO: 15, hSec61β с последовательностью SEQ ID NO: 17, hSec61γ с последовательностью SEQ ID NO: 19, hSRP54 с последовательностью SEQ ID NO: 21, hSRP9 с последовательностью SEQ ID NO: 23, hSRPRα с последовательностью SEQ ID NO: 25, hSRPβ с последовательностью SEQ ID NO: 27 и hCANX с последовательностью SEQ ID NO: 29, и/или может соответствовать последовательностям аминокислот, идентичным более чем на 80%, 90%, 95% или 98% указанным последовательностям.
Белок ТЕР может представлять собой один или более из следующих белков процессинга белка или метаболических белков: hUCP4, hCMPSAT, rST6Gal1, hCOMSC, T-синтазу человека, hP4HA1, hP4HB, hGILZ, hCyPB, hNRF2, hHK1, hPDI, hPIN1, hSEPW1, hCALR, hDDOST, hHSP40, hATP5A1, hSERCA2, hPDIA4, hHSC70/HSPA8, hHYOU1, hCMP-SAS, беклин-1 человека, hERdj3, CHO-AGE, hWip1, hRTP4, hREEP2, hDPM1 и hDRiP78.
Белок ТЕР также может соответствовать одной или более из следующих последовательностей аминокислот белков процессинга белка или метаболических белков: hUCP4 с последовательностью SEQ ID NO: 31, hCMPSAT с последовательностью SEQ ID NO: 33, rST6Gal1 с последовательностью SEQ ID NO: 35, hCOMSC с последовательностью SEQ ID NO: 37, Т-синтазы человека с последовательностью SEQ ID NO: 39, hP4HA1 с последовательностью SEQ ID NO: 41, hP4HB с последовательностью SEQ ID NO: 43, hGILZ с последовательностью SEQ ID NO: 45, hCyPB с последовательностью SEQ ID NO: 47, hNRF2 с последовательностью SEQ ID NO: 49, hHK1 с последовательностью SEQ ID NO: 51, hPDI с последовательностью SEQ ID NO: 53, hPIN1 с последовательностью SEQ ID NO: 55, hSEPW1 с последовательностью SEQ ID NO: 57, hCALR с последовательностью SEQ ID NO: 59, hDDOST с последовательностью SEQ ID NO: 62, hHSP40 с последовательностью SEQ ID NO: 64, hATP5A1 с последовательностью SEQ ID NO: 66, hSERCA2 с последовательностью SEQ ID NO: 68, hPDIA4 с последовательностью SEQ ID NO: 70, hHSC70/HSPA8 с последовательностью SEQ ID NO: 72, hHYOU1 с последовательностью SEQ ID NO: 74, hCMP-SAS с последовательностью SEQ ID NO: 76, беклина-1 человека с последовательностью SEQ ID NO: 78, hERdj3 с последовательностью SEQ ID NO: 80, CHO-AGE с последовательностью SEQ ID NO: 82, hWip1 с последовательностью SEQ ID NO: 84, hRTP4 с последовательностью SEQ ID NO: 86, hREEP2 с последовательностью SEQ ID NO: 88, hDPM1 с последовательностью SEQ ID NO: 90 и hDRiP78 с последовательностью SEQ ID NO: 92, и/или может соответствовать последовательностям аминокислот, идентичным более чем на 80%, 90%, 95% или 98% указанным последовательностям.
Белок ТЕР может представлять собой шаперон, в частности, белок BiP, а конкретнее, сконструированное производное белка BiP дрозофилы (DroBiP), последовательность которого на 80%, 90%, 95% или 100% идентична последовательности SEQ ID NO: 60.
Функциональная РНК ТЕР внутри рекомбинантной клетки млекопитающего может включать/состоять из последовательности нуклеиновой кислоты (последовательностей нуклеиновых кислот), кодирующей функциональную РНК, предпочтительно микроРНК или кшРНК, которая препятствует экспрессии по меньшей мере одного белка рекомбинации, предпочтительно гена гомологичной рекомбинации, такого как, но не ограничиваясь перечисленными: Rad51, Rad51B, Rad51C, Rad51D, Xrcc2, Xrcc3, Rad52, Rad54, Brca1, Brca2, циклин D1, Ercc1, MDC1, Bard1, лигаза 1, Mre11 и/или 53BP1. Последовательность нуклеиновых кислот может быть по меньшей мере на 80%, 90%, 95%, 98% или 100% идентична Rad51 с последовательностью SEQ ID NO: 93, Rad51B с последовательностью SEQ ID NO: 94, Rad51C с последовательностью SEQ ID NO: 95, Rad51D с последовательностью SEQ ID NO: 96, Xrcc2 с последовательностью SEQ ID NO: 99, Xrcc3 с последовательностью SEQ ID NO: 100, Rad52 с последовательностью SEQ ID NO: 97, Rad54 с последовательностью SEQ ID NO: 98, Brcal с последовательностью SEQ ID NO: 101, Brca2 с последовательностью SEQ ID NO: 102, циклину D1 с последовательностью SEQ ID NO: 103, Ercc1 с последовательностью SEQ ID NO: 104, MDC1 с последовательностью SEQ ID NO: 105, Bard1 с последовательностью SEQ ID NO: 106, лигазе 1 с последовательностью SEQ ID NO: 107, Mre11 с последовательностью SEQ ID NO: 108 и/или 53BP1 с последовательностью SEQ ID NO: 109.
Рекомбинантная клетка млекопитающего может представлять собой первичную стволовую клетку, клетку хомячка, например, клетку яичника китайского хомячка (СНО), или клетку человека, например, клетку HEK293.
Настоящее изобретение также направлено на рекомбинантную клетку млекопитающего, содержащую:
a.) по меньшей мере одну функциональную РНК ТЕР и/или по меньшей мере одну рекомбинантную последовательность нуклеиновой кислоты, кодирующую белок ТЕР или кодирующую функциональную РНК ТЕР,
и
b.) рекомбинантную молекулу нуклеиновой кислоты, содержащую:
(i) по меньшей мере один интересующий трансген, и
(ii) возможно, элемент MAR.
Выше отмечено, что белок ТЕР может представлять собой один или более из следующих белков пути секреции белка: hSRP14, hSec61α1, hSec61β, hSec61γ, hSRP54, hSRP9, hSRPRα, hSRPβ и hCANX.
Белок ТЕР также может соответствовать одной или более из следующих последовательностей аминокислот белков пути секреции белка: hSRP14 с последовательностью SEQ ID NO: 13, hSec61α1 с последовательностью SEQ ID NO: 15, hSec61β с последовательностью SEQ ID NO: 17, hSec61γ с последовательностью SEQ ID NO: 19, hSRP54 с последовательностью SEQ ID NO: 21, hSRP9 с последовательностью SEQ ID NO: 23, hSRPRα с последовательностью SEQ ID NO: 25, hSRPβ с последовательностью SEQ ID NO: 27, и hCANX с последовательностью SEQ ID NO: 29 и/или может соответствовать последовательностям аминокислот, идентичным более чем на 80%, 90%, 95% или 98% указанным последовательностям.
Белок ТЕР может представлять собой один или более из следующих белков процессинга белка или метаболических белков: hUCP4, hCMPSAT, rST6GaI1, hCOMSC, T-синтазу человека, hP4HA1, hP4HB, hGILZ, hCyPB, hNRF2, hHK1, hPDI, hPIN1, hSEPW1, hCALR, hDDOST, hHSP40, hATP5A1, hSERCA2, hPDIA4, hHSC70/HSPA8, hHYOU1, hCMP-SAS, беклин-1 человека, hERdj3, CHO-AGE, hWip1, hRTP4, hREEP2, hDPM1 и hDRiP78.
Белок ТЕР также может соответствовать одной или более из следующих последовательностей аминокислот белков процессинга белка или метаболических белков: hUCP4 с последовательностью SEQ ID NO: 31, hCMPSAT с последовательностью SEQ ID NO: 33, rST6GaI1 с последовательностью SEQ ID NO: 35, hCOMSC с последовательностью SEQ ID NO: 37, Т-синтазы человека с последовательностью SEQ ID NO: 39, hP4HA1 с последовательностью SEQ ID NO: 41, hP4HB с последовательностью SEQ ID NO: 43, hGILZ с последовательностью SEQ ID NO: 45, hCyPB с последовательностью SEQ ID NO: 47, hNRF2 с последовательностью SEQ ID NO: 49, hHK1 с последовательностью SEQ ID NO: 51, hPDI с последовательностью SEQ ID NO: 53, hPIN1 с последовательностью SEQ ID NO: 55, hSEPW1 с последовательностью SEQ ID NO: 57, hCALR с последовательностью SEQ ID NO: 59, hDDOST с последовательностью SEQ ID NO: 62, hHSP40 с последовательностью SEQ ID NO: 64, hATP5A1 с последовательностью SEQ ID NO: 66, hSERCA2 с последовательностью SEQ ID NO: 68, hPDIA4 с последовательностью SEQ ID NO: 70, hHSC70/HSPA8 с последовательностью SEQ ID NO: 72, hHYOU1 с последовательностью SEQ ID NO: 74, hCMP-SAS с последовательностью SEQ ID NO: 76, беклина-1 человека с последовательностью SEQ ID NO: 78, hERdj3 с последовательностью SEQ ID NO: 80, CHO-AGE с последовательностью SEQ ID NO: 82, hWip1 с последовательностью SEQ ID NO: 84, hRTP4 с последовательностью SEQ ID NO: 86, hREEP2 с последовательностью SEQ ID NO: 88, hDPM1 с последовательностью SEQ ID NO: 90 и hDRiP78 с последовательностью SEQ ID NO: 92 и/или может соответствовать последовательностям аминокислот, идентичным более чем на 80%, 90%, 95% или 98% указанным последовательностям.
Белок ТЕР может представлять собой шаперон, в частности, белок BiP, а конкретнее, синтетическое производное белка BiP дрозофилы (DroBiP), последовательность которого на 80%, 90%, 95% или 100% идентична последовательности SEQ ID NO: 60.
Функциональная РНК может представлять собой временно трансфицированную миРНК или кшРНК, которая транскрибируется с указанной по меньшей мере одной изолированной последовательности нуклеиновой кислоты, при этом указанная миРНК или указанная процессированная кшРНК представляет собой антисмысловую РНК длиной 20, 21, 22, 23, 24 или 25 пар оснований, которая полностью комплементарна 20, 21, 22, 23, 24 или 25 последовательным нуклеотидам мРНК по меньшей мере одного целевого гена, который является частью рекомбинантного пути НГСК (негомологичного соединения концов), ГР (гомологичной рекомбинации), ОМСК (опосредованного микрогомологией соединения концов) или представляет собой белок репарации ДНК, такой как MDC1 (медиатор контрольных точек повреждения ДНК 1).
Указанный по меньшей мере один целевой ген, который может быть частью:
- репарации ДНК и НГСК, представляет собой
53ВР1 (белок 1, связывающий опухолевый супрессор р53),
- ГР, представляет собой
Rad51 (белок репарации ДНК RAD51), Rad51B (гомолог 2 белка репарации ДНК RAD51), Rad51C (гомолог 3 белка репарации ДНК RAD51),
Rad51D (гомолог 4 белка репарации ДНК RAD51), Rad52 (белок репарации ДНК RAD52), Rad54 (белок репарации и рекомбинации ДНК RAD54),
Xrcc2 (репарация после облучения рентгеновскими лучами, исправляющая (комплементирующая) нарушенную репарацию в клетках китайского хомячка 2),
Xrcc3 (репарация после облучения рентгеновскими лучами, комплементирующая нарушенную репарацию в клетках китайского хомячка 3),
Brca1 (рак молочной железы 1, раннее начало),
Brca2 (рак молочной железы 2, раннее начало),
Bard1 (ассоциированный с BRCA1 RING-домен 1),
- ОМСК, представляет собой
Ercc1 (эксцизионная репарация, кросс-комплементирующая нарушенную репарацию у грызунов, группа комплементации 1),
Mre11 (мейотическая рекомбинация 11)
лигазу 1 (ДНК-лигаза 1),
и/или
представляет собой белок репарации ДНК MDC1.
Целевые гены могут представлять собой нуклеиновые кислоты, последовательность которых по меньшей мере на 80%, 90%, 95%, 98% или 100% идентична Rad51 с последовательностью SEQ ID NO: 93, Rad51B с последовательностью SEQ ID NO: 94, Rad51C с последовательностью SEQ ID NO: 95, Rad51D с последовательностью SEQ ID NO: 96, Xrcc2 с последовательностью SEQ ID NO: 99, Xrcc3 с последовательностью SEQ ID NO: 100, Rad52 с последовательностью SEQ ID NO: 97, Rad54 с последовательностью SEQ ID NO: 98, Brca1 с последовательностью SEQ ID NO: 101, Brca2 с последовательностью SEQ ID NO: 102, циклину D1 с последовательностью SEQ ID NO: 103, Ercc1 с последовательностью SEQ ID NO: 104, MDC1 с последовательностью SEQ ID NO: 105, Bard1 с последовательностью SEQ ID NO: 106, лигазе 1 с последовательностью SEQ ID NO: 107, Mre11 с последовательностью SEQ ID NO: 108 и/или 53BP1 с последовательностью SEQ ID NO: 109.
С указанного по меньшей мере одного трансгена можно экспрессировать терапевтический белок, такой как иммуноглобулин, гормон, такой как эритропоэтин, или фактор роста, и при этом необязательно встраивание и/или экспрессия трансгена повышена в рекомбинантной клетке млекопитающего по сравнению с клеткой, не содержащей указанную рекомбинантную молекулу(ы) нуклеиновой кислоты.
Рекомбинантная клетка млекопитающего может содержать по меньшей мере две функциональные РНК ТЕР, при этом одна или обе РНК ТЕР представляют собой временно трансфицированные миРНК, или экспрессируются с указанной изолированной последовательности(ей) нуклеиновой кислоты, кодирующей функциональную РНК ТЕР.
Рекомбинантная клетка млекопитающего может содержать элемент MAR.
Настоящее изобретение также направлено на способ трансфекции клеток млекопитающих, в частности, клеток хомячка, включающий:
трансфекцию, возможно при первой трансфекции, указанных клеток млекопитающих
(i) по меньшей мере одной из указанных рекомбинантных молекул нуклеиновых кислот по любому из пп. 1-13 и/или
(ii) по меньшей мере одной изолированной функциональной РНК ТЕР и по меньшей мере одним трансгеном, который необязательно является частью рекомбинантной молекулы нуклеиновой кислоты, которой возможно трансфицируют при второй, последующей трансфекции, возможно вместе с изолированной нуклеиновой кислотой или мРНК, экспрессирующей транспозазу, которая узнает указанные 5'- и 3'-ITR.
Рекомбинантную клетку млекопитающего можно трансфицировать более чем одной молекулой нуклеиновой кислоты, включая по меньшей мере две, по меньшей мере три или по меньшей мере четыре из указанных рекомбинантных молекул нуклеиновых кислот, кодирующих один, два или три из следующих белков: hSRP14, hSec61α1, hSec61β, hSec61γ, hSRP54, hSRP9, hSRPRα, hSRPβ и hCANX.
Ее также можно трансфицировать hSRP14 с последовательностью SEQ ID NO: 13, hSec61α1 с последовательностью SEQ ID NO: 15, hSec61β с последовательностью SEQ ID NO: 17, hSec61γ с последовательностью SEQ ID NO: 19, hSRP54 с последовательностью SEQ ID NO: 21, hSRP9 с последовательностью SEQ ID NO: 23, hSRPRα с последовательностью SEQ ID NO: 25, hSRPβ с последовательностью SEQ ID NO: 27, и hCANX с последовательностью SEQ ID NO: 29, и/или последовательностями аминокислот, идентичными более чем на 80%, 90%, 95% или 98% указанным последовательностям.
Любые из рекомбинантных молекул нуклеиновых кислот могут представлять собой часть вектора, при этом можно осуществить котрансфекцию указанными векторами.
Котрансфекция векторами, кодирующими несколько белков ТЕР, предпочтительно котрансфекция рекомбинантными молекулами нуклеиновых кислот, кодирующими белки SRP14, SRP9 и SRP54, может повышать встраивание и/или экспрессию трансгена в указанной клетке по сравнению с клеткой, не подвергнутой такой котрансфекции. Котрансфекция векторами, содержащими последовательности нуклеиновых кислот, которые по меньшей мере на 80%, 90%, 95%, 98% или 100% идентичны последовательностям SEQ ID NO: 12, SEQ ID NO: 22, SEQ ID NO: 20, также входит в объем настоящего изобретения.
Можно получить множество указанных клеток млекопитающих, которые стабильно экспрессируют указанный белок ТЕР или функциональную РНК ТЕР, чтобы получить рекомбинантные клетки млекопитающих, и при этом указанное количество рекомбинантных клеток млекопитающих может не зависеть от наличия указанного элемента MAR. Указанные клетки млекопитающих можно трансфицировать два и возможно три раза.
Предпочтительно, по меньшей мере 30%, 40% или 45% указанных клеток млекопитающих могут стать рекомбинантными клетками млекопитающих и экспрессировать указанный трансген.
Клетку млекопитающего можно трансфицировать указанной по меньшей мере одной изолированной функциональной РНК ТЕР и вектором, содержащим указанный по меньшей мере один трансген и необязательно 3'-ITR и 5'-ITR, фланкирующие указанный по меньшей мере один трансген, возможно вместе с изолированной нуклеиновой кислотой или мРНК, экспрессирующей транспозазу, которая узнает указанные 5'- и 3'-ITR.
Трансген может представлять собой терапевтический белок, такой как иммуноглобулин, гормон, цитокин или фактор роста.
Рекомбинантная молекула нуклеиновой кислоты может необязательно содержать маркер отбора, и при этом указанный по меньшей мере один трансген экспрессируется
(а) без отбора по указанному маркеру, или
(b) с отбором по указанному маркеру, например, путем добавления селекционного агента в культуральную среду, и
(c) в отсутствие транспозазы, или
(d) в присутствии транспозазы.
Функциональная РНК ТЕР может кодироваться рекомбинантными последовательностями нуклеиновых кислот, кодирующими кшРНК или микроРНК, или может включать/состоять из миРНК, которая препятствует экспрессии по меньшей мере одного гена гомологичной рекомбинации, такого как, но не ограничиваясь перечисленными: Rad51, Rad51B, Rad51C, Rad51D, Xrcc2, Xrcc3, Rad52, Rad54, Brca1, Brca2, циклин D1, Ercc1, MDC1, Bardl, лигаза 1, Mre11 и/или 53BP1.
Функциональная РНК ТЕР также может препятствовать экспрессии генов, последовательность которых по меньшей мере на 80%, 90%, 95%, 98% или 100% идентична Rad51 с последовательностью SEQ ID NO: 93, Rad51B с последовательностью SEQ ID NO: 94, Rad51C с последовательностью SEQ ID NO: 95, Rad51D с последовательностью SEQ ID NO: 96, Xrcc2 с последовательностью SEQ ID NO: 99, Xrcc3 с последовательностью SEQ ID NO: 100, Rad52 с последовательностью SEQ ID NO: 97, Rad54 с последовательностью SEQ ID NO: 98, Brca1 с последовательностью SEQ ID NO: 101, Brca2 с последовательностью SEQ ID NO: 102, циклину D1 с последовательностью SEQ ID NO: 103, Ercc1 с последовательностью SEQ ID NO: 104, MDC1 с последовательностью SEQ ID NO: 105, Bardl с последовательностью SEQ ID NO: 106, лигазе 1 с последовательностью SEQ ID NO: 107, Mre11 с последовательностью SEQ ID NO: 108 и/или 53BP1 с последовательностью SEQ ID NO: 109.
Встраивание и/или экспрессию трансгена можно повысить в такой клетке по сравнению с клеткой, не трансфицированной указанными изолированными молекулами нуклеиновых кислот и/или по меньшей мере одной из указанных изолированных функциональных РНК ТЕР.
Настоящее изобретение также направлено на набор, в одном контейнере содержащий по меньшей мере один вектор, содержащий любую из указанных рекомбинантных молекул нуклеиновых кислот согласно пп. 1-13, и во втором необязательном контейнере содержащий вектор, кодирующий подходящую транспозазу, и в дополнительном контейнере содержащий инструкцию по применению указанного вектора или векторов.
Предложен указанный выше набор, в котором более чем один вектор предусмотрен в одном или более контейнерах и в котором белки ТЕР представляют собой по меньшей мере два из следующих белков: шаперон, SRP14, SRP9, SRP54, SR или транслокон.
Предложен указанный выше набор, в котором функциональная(ые) РНК ТЕР внутри указанного вектора(ов) включает(ют)/состоит(ят) из последовательностей нуклеиновых кислот, кодирующих микроРНК, миРНК или кшРНК, которые препятствуют экспрессии по меньшей мере одного гена гомологичной рекомбинации, такого как, но не ограничиваясь перечисленными: Rad51, Rad51B, Rad51C, Rad51D, Хгсс2, Xrcc3, Rad52, Rad54, Brca1, Brca2, циклин D1, Ercc1, MDC1, Bard1, лигаза 1, Mre11 и/или 53BP1, и предпочтительно в дополнительном контейнере указанный набор содержит одну или более миРНК, которые препятствуют экспрессии по меньшей мере одного другого гена гомологичной рекомбинации, такого как, но не ограничиваясь перечисленными: Rad51, Rad51B, Rad51C, Rad51D, Xrcc2, Xrcc3, Rad52, Rad54, Brca1, Brca2, циклин D1, Ercc1, MDC1, Bard1, лигаза 1, Mre11 и/или 53BP1.
Ген гомологичной рекомбинации может соответствовать нуклеиновым кислотам, последовательность которых по меньшей мере на 80%, 90%, 95%, 98% или 100% идентична Rad51 с последовательностью SEQ ID NO: 93, Rad51B с последовательностью SEQ ID NO: 94, Rad51C с последовательностью SEQ ID NO: 95, Rad51D с последовательностью SEQ ID NO: 96, Xrcc2 с последовательностью SEQ ID NO: 99, Xrcc3 с последовательностью SEQ ID NO: 100, Rad52 с последовательностью SEQ ID NO: 97, Rad54 с последовательностью SEQ ID NO: 98, Brca1 с последовательностью SEQ ID NO: 101, Brca2 с последовательностью SEQ ID NO: 102, циклину D1 с последовательностью SEQ ID NO: 103, Ercc1 с последовательностью SEQ ID NO: 104, MDC1 с последовательностью SEQ ID NO: 105, Bard1 с последовательностью SEQ ID NO: 106, лигазе 1 с последовательностью SEQ ID NO: 107, Mre11 с последовательностью SEQ ID NO: 108 и/или 53BP1 с последовательностью SEQ ID NO: 109.
Настоящее изобретение также направлено на применение рекомбинантных нуклеиновых кислот, описанных в данной заявке, и/или рекомбинантных клеток млекопитающих, описанных в данной заявке, предпочтительно для повышения встраивания и/или экспрессии трансгена.
Настоящее изобретение также направлено на вектор экспрессии, содержащий:
(a) трансген, который фланкирован в 3'-5' направлении промотором и в 5'-3' направлении сигналом полиаденилирования,
(b) единичный элемент MAR в 5'-3' направлении от сигнала полиаденилирования, или
(c) первый элемент MAR в 3'-5' направлении от интересующего трансгена и второй элемент MAR в 5'-3' направлении от места встраивания указанного трансгена.
Единичный или первый и второй элементы MAR можно выбрать из элементов MAR 1_68, 1_6, 1_6R2, 1_68R, 1_68R2, X_29R3 или X_29 или элементов, последовательности которых по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентичны последовательностям SEQ ID NO: 1, 2, 3, 6, 7, 8, 9 или 10.
Единичный или первый и второй элемент(ы) MAR можно выбрать из перестроенных элементов MAR 1_6R2, 1_68R, 1_68R2 или X_29R3 или элементов, последовательности которых по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентичны последовательностям SEQ ID NO: 6, 7, 8, или 10, при этом необязательно указанный элемент(ы) MAR повышает экспрессию интересующего трансгена на по меньшей мере 10%, по меньшей мере 20%, по меньшей мере 30%, по меньшей мере 40%, по меньшей мере 50%, по меньшей мере 60% или по меньшей мере 70% по сравнению с их неперестроенными аналогами.
Промотор может представлять собой промотор EF1-альфа и сигнал полиаденилирования представляет собой поли(А) сигнал BGH.
Указанный вектор может содержать промотор(ы) и/или энхансер(ы) или продукты их слияния, такие как GAPDH, CGAPD, CSV40p, CMVp, СНО Ef1-альфа, Actb СНО или Hspa5 СНО.
Промоторы могут представлять собой промоторы GAPDH с последовательностью SEQ ID NO: 111, CGAPDH с последовательностью SEQ ID NO: 11, SV40p с последовательностью SEQ ID NO: 114, CMVp с последовательностью SEQ ID NO: 113, EF1-альфа СНО с последовательностью SEQ ID NO: 112, Actb СНО с последовательностью SEQ ID NO: 115 и/или Hspa5 СНО с последовательностью SEQ ID NO: 116 и последовательности нуклеиновых кислот, идентичные более чем на 80%, 90%, 95% или 98% указанным последовательностям.
Промотор может представлять собой промотор GAPDH и включать энхансер CMV.
Первый и/или второй MAR, энхансер, промотор, интересующий трансген и сигнал полиаденилирования могут быть расположены между 5'- и 3'-ITR.
В некотором варианте реализации указанный вектор экспрессии может содержать:
(a) единичный элемент MAR в 5'-3' направлении от сигнала полиаденилирования, при этом указанный единичный элемент MAR предпочтительно представляет собой элемент MAR 1-68 или MARX-29 и/или элемент, последовательность которого по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентична последовательности SEQ ID NO: 1 или 3, в частности, перестроенный MAR на основе MAR 1-68 или MAR Х-29, в частности, элемент, последовательность которого по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентична последовательностям SEQ ID NO: 6, 7 или 10 (элементы MAR 1_68R, 1_68R2 или X_29R3) или SEQ ID NO: 9, и предпочтительно представляет собой элемент MAR Х-29, и/или его последовательность по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентична последовательности SEQ ID NO: 3, или
(b) первый элемент MAR в 3'-5' направлении от интересующего трансгена и второй элемент MAR в 5'-3' направлении от указанного интересующего трансгена, при этом указанный первый элемент MAR предпочтительно включает элемент 1_6, и/или его последовательность по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентична последовательности SEQ ID NO: 2, в частности, идентична перестроенным элементам MAR на основе MAR 1-6, в частности, элементам, последовательности которых по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентичны последовательности SEQ ID NO: 8 (MAR 1_6R2), и второй элемент MAR, который предпочтительно включает элемент MAR 1-68, и/или его последовательность по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентична последовательности SEQ ID NO: 1.
Вектор экспрессии может содержать единичный элемент MAR, и указанный единичный элемент MAR может быть расположен в 5'-3' направлении от сайта полиаденилирования и представляет собой MAR 1-68 или MAR Х-29, и/или его последовательность по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентична последовательности SEQ ID NO: 1 или 3, в частности, идентична перестроенным элементам MAR на основе MAR 1-68 или MAR Х-29, в частности, элементам, последовательность которых по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентична последовательностям SEQ ID NO: 6, 7 или 10 (элементы MAR 1_68R, 1_68R2 или X_29R3) или 9, и может предпочтительно представлять собой полученный из MAR Х-29 элемент, и/или его последовательность по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентична SEQ ID NO: 3.
Указанный первый элемент MAR может находиться в 3'-5' направлении от интересующего трансгена и второй элемент MAR может находиться в 5'-3' направлении от указанного интересующего трансгена, при этом указанный первый элемент MAR может включать элемент MAR 1_6, и/или его последовательность по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентична последовательности SEQ ID NO 2, и указанный второй элемент MAR может включать элемент MAR 1_68, и/или его последовательность может быть по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентична SEQ ID NO: 1.
Настоящее изобретение также направлено на способ экспрессии трансгена, включающий:
обеспечение рекомбинантной клетки млекопитающего, содержащей один из упомянутых выше векторов, содержащих указанный трансген и экспрессирующих указанный трансген, при этом указанный элемент(ы) MAR может повышать экспрессию указанного трансгена предпочтительно на по меньшей мере 10%, по меньшей мере 20%, по меньшей мере 30%, по меньшей мере 40%, по меньшей мере 50%, по меньшей мере 60% или по меньшей мере 70%. Указанный вектор может содержать единичный элемент MAR Х_29 и/или нуклеиновую кислоту, последовательность которой по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентична последовательности SEQ ID NO: 3, и при этом через более чем 4, 5, 6, 7, 8, 9, 10, 11, 12, 13 или 14 недель культивирования указанный элемент MAR может повышать экспрессию интересующего трансгена на по меньшей мере 10%, по меньшей мере 20%, по меньшей мере 30%, по меньшей мере 40%, по меньшей мере 50%, по меньшей мере 60% или по меньшей мере 70%.
КРАТКОЕ ОПИСАНИЕ ЧЕРТЕЖЕЙ
Фиг. 1. Конструирование вектора, несущего транспозон.
Для того, чтобы проверить, может ли влиять добавление элемента MAR к транспозону РВ (PiggyBac) на эффективность транспозиции и экспрессию трансгена, и чтобы оценить, оказывало ли расположение MAR в конструкции какое-либо влияние на данные эффекты, разработали серию донорных конструкций транспозонов, содержащих ген зеленого флуоресцентного белка (GFP) и ген устойчивости к пуромицину (Puro), в которые встроили MAR 1_68 или контрольную нейтральную спейсерную последовательность ДНК в различные положения в указанной плазмиде. Исходную плазмиду, несущую транспозон, Puro-GFP без вставки использовали в качестве контроля транспозиции, чтобы отличить вклад увеличенного размера транспозона от влияния добавления MAR или спейсерной последовательности.
Фиг. 2. Векторы, несущие транспозоны: эффективность транспозиции.
Эффективность транспозиции различных конструкций, несущих транспозоны, измеряли путем оценки (А) процента клеток, экспрессирующих GFP, после трансфекции и трех недель культивирования без селекции антибиотиком и (В) путем подсчета устойчивых к пуромицину колоний.
Фиг. 3. Векторы, несущие транспозоны: уровень экспрессии.
Анализ уровня экспрессии, обеспеченного различными векторами, несущими транспозоны, трансфицированными совместно (+РВ) или без (-РВ) плазмиды, экспрессирующей транспозазу, путем измерения уровней флуоресценции GFP в клетках СНО после 3 недель культивирования без селекции (А) или при селекции (В) по устойчивости к пуромицину после трансфекции, с учетом флуоресценции только GFP-положительных клеток.
Фиг. 4. Влияние MAR и транспозазы на встраивание трансгена в геном.
Количество встроенных копий GFP-трансгена определяли, применяя количественную ПЦР, и значения нормировали по отношению к клеточному гену В2М, применяя геномную ДНК, выделенную из не подвергнутых селекции клеток СНО (А), или устойчивых к пуромицину клеток (В), полученных как описано в подписях к фигурам 1-3. Значения представляют средние значения ± стандартная ошибка среднего (n=3). *Р<0,05.
Фиг. 5. Экспрессия трансгена относительно трансгена.
Оценка внутреннего экспрессионного потенциала векторов независимо от их способности встраиваться в геном, без селекции (А) и при селекции (В) пуромицином.
Фиг. 6. Влияние экспрессии секреторных белков с векторов на основе транспозонов и плазмидных векторов на экспрессию рекомбинантного белка (трансгена). Векторы на основе транспозонов или обычные плазмидные векторы сконструировали для экспрессии секреторных белков SRP9, SRP14, SRP54, альфа и бета субъединиц (SR) рецептора SRP или транслокона. Векторы на основе транспозонов котрансфицировали с вектором экспрессии транспозазы PiggyBac (правая панель), тогда как не основанные на транспозонах плазмидные векторы трансфицировали отдельно (левая панель) в клон клетки, экспрессирующей антитело инфликсимаб, как описано в данном тексте. После трех недель культивирования при селекции (левая панель) или без селекции (правая панель) анализировали уровни секретируемого антитела инфликсимаба в культуральных супернатантах. Можно увидеть, что удельная продуктивность, которая представляет собой относительную экспрессию в клетке, содержащей последовательность, кодирующую белок экспрессии/процессинга трансгена (ТЕР), или функциональную РНК ТЕР, повышалась при применении вектора, несущего транспозон, от 0,25-1,5 до 1-2,5, соответственно, по сравнению с исходной клеткой без ТЕР.
Фиг. 7. Экспрессия рекомбинантного белка в суспензиях подвергнутых электропорации клеток СНО-М.
(A) Клетки СНО-М, которые подвергали электропорации один или два раза несущим MAR Х-29 вектором, экспрессирующим GFP и транспозон, в присутствии (+РВ) или отсутствие (-РВ) транспозазы piggyBac. Показан процент стабильно экспрессирующих GFP клеток после 3 недель культивирования, осуществленного в отсутствие селекции.
(B) Среднее значение флуоресценции GFP в GFP-положительных клетках.
(C) кДНК, кодирующие легкие и тяжелые цепи антитела бевацизумаба (Beva), адалимумаба (Adal) и ритуксимаба (Ritu), встраивали вместо GFP в плазмиды, несущие транспозон, содержащие MAR Х29. Клетки СНО-М подвергали электропорации три раза с интервалами по 12 дней конструкциями, несущими транспозоны, содержащими легкую и тяжелую цепь, вместе с вектором экспрессии, несущим транспозазу piggyBac. Показаны уровни иммуноглобулина, секретируемого в культуральные супернатанты совокупностью поликлональных клеток, которых растили без селекции (незакрашенные столбики). В качестве альтернативы, не подвергнутые селекции поликлональные популяции клеток подвергали сортировке путем пэннинга (просеивания) клеток, на поверхностях которых были выставлены иммуноглобулины, применяя магнитные микрогранулы. Показаны уровни иммуноглобулинов, секретируемых не подвергнутыми сортировке популяциями (закрашенные столбики).
(D) Популяции трансфицированных клеток подвергали сортировке для обнаружения экспрессирующих иммуноглобулины колоний, применяя устройство, собирающее колонии, и по два клона, экспрессирующих каждый из трех указанных иммуноглобулинов, растили в культурах с подпиткой в биореакторах в виде центрифужных пробирок. Показаны уровни секретируемых иммуноглобулинов, и их определяли также, как и для панели (С).
Фиг. 8. Гетерологичная экспрессия SRP14 улучшает секрецию трастузумаба и восстанавливает секрецию инфликсимаба.
Клоны HP и LP СНО-K1, экспрессирующие иммуноглобулины трастузумаб (А) или инфликсимаб (В) на наиболее высоких из полученных уровней, повторно стабильно трансфицировали вектором экспрессии SRP14, и выделяли моноклональные популяции. Оценивали рост и продуктивность клеток в условиях периодической культуры для полученных субклонов, обозначенных от А до Е. Концентрацию клеток (клетки/мл) и титр IgG (мкг/мл) наносили на график для каждого дня отбора образцов на протяжении 7 дней культивирования. (С) Распределение удельной продуктивности субклонов трастузумаба (HP) и инфликсимаба (LP) после трансфекции вектором экспрессии SRP14 (дорожки S) по сравнению с таковой для исходных клонов HP и LP (-). (D) Определяли относительные уровни мРНК SRP14 для 5 отдельных субклонов А-Е SRP14-LP и исходного контрольного клона LP, и наносили их на график в зависимости от удельной продуктивности IgG для 4 полученных культур. Средние значения и стандартные отклонения уровней мРНК и удельной продуктивности выражали в виде кратности превышения соответствующих значений для контрольного клона LP.
Фиг. 9. Гетерологичная экспрессия SRP14 опосредует высокий выход с трудом экспрессируемого иммуноглобулина в процессе получения.
Трансфицированные вектором, несущим SRP14, субклон HP В, экспрессирующий трастузумаб (А), и субклон LP Е, экспрессирующий инфликсимаб (В), проанализированные на фиг. 8, культивировали в вентилируемых встряхиваемых колбах объемом 125 мл в культурах с подпиткой с рабочим объемом 25 мл и определяли концентрацию жизнеспособных клеток и титр IgG в течение 11-дневного периода.
Фиг. 10. Экспрессия SRP14 нарушает агрегацию легких цепей в клонах клеток СНО.
(А) Собранные путем центрифугирования супернатанты и осадки пермеабилизованных Triton Х100 (Tx100) клеток анализировали с помощью ЭФ в ПААГ/ДСН, как изображено на панелях с обозначением растворимых в Тх-100 и нерастворимых в Тх-100 агрегатов, соответственно, для субклона Е SRP14-LP, полученного из LP, и субклона В SRP14-HP, полученного из HP (дорожки S), или для субклонов СНО, экспрессирующих контрольный белок GFP (дорожки G). Стрелками показаны недостаточно процессированная свободная легкая цепь (LC) и агрегировавшая (агр.) LC. (В) Осуществляли анализ с вытеснением для различных промежуточных вариантов агрегации LC, НС и IgG, полученных в клоне Е SRP14-LP и LP-контрольном клоне Е, результаты которого показаны на данной фигуре.
Фиг. 11. Влияние совместной экспрессии субъединиц SRP, SR и транслокона на секрецию иммуноглобулина.
(А) Клон LP Е, экспрессирующий инфликсимаб, повторно трансфицировали различными комбинациями векторов экспрессии на основе транспозонов, несущих SRP, SR и транслокон. Удельную продуктивность полученных популяций клеток затем оценивали при периодическом культивировании и представили в виде % от значений в пикограммах на клетку в день (pcd) для LP-контрольных клеток. На диаграммах размаха представлены средняя, верхняя и нижняя квартили нормированных удельных продуктивностей, определенных в день 3 для независимых серий культивирования. (В) Субклон клетки Е, экспрессирующий SRP14 и продуцирующий инфликсимаб, повторно трансфицировали различными комбинациями векторов экспрессии на основе транспозонов, несущих SR и транслокон. Удельная продуктивность популяций клеток представлена аналогично панели А.
Фиг. 12. Модель восстановления секреции инфликсимаба экспрессирующими SRP14 клонами
Модель фолдинга и секреции IgG клонами с низкой продуцирующей способностью до (А) и после (В) сверхэкспрессии субъединиц SRP/транслокона. Полученные результаты свидетельствуют о том, что вновь синтезированная LC, продуцируемая клонами с низкой продуцирующей способностью, недостаточно процессируется и имеет неправильный фолдинг. Недостаточный процессинг сигнального пептида LC инфликсимаба может приводить к насыщению аппарата котрансляционной транслокации ЭР (панель А, номер 1). Ее агрегация в ЭР внутри не способных к сборке IgG агрегировавших форм LC (панель А, номер 2) вызывает ЭР-стресс и вызывает образование подобной аутофагосоме структуры (панель А, номер 3). Сверхэкспрессия SRP14 и других компонентов белков SRP/транслокона полностью восстанавливала процессинг и секрецию инфликсимаба IgG (панель В). Активность SRP14, останавливающая элонгацию, возможно задерживает перемещение LC в ЭР в процессе трансляции ее мРНК (панель В, номер 1). Это, в свою очередь, будет способствовать правильному процессингу LC и ее правильному взаимодействию с осуществляющими фолдинг шаперонами в ЭР (панель В, номер 2). Поддержание вновь синтезированной LC в состоянии, позволяющем сборку IgG, таким образом, восстанавливает высокий выход секреции полностью собранных антител (панель В, номер 3).
Фиг. 13. Влияние подавления ГР и НГСК молекулами миРНК на экспрессию.
Кратность различий в проценте GFP-положительных клеток (по сравнению с клетками, трансфицированными контрольной плазмидой GFP, представленными на данной фигуре как 1,0), представляющем частоты событий рекомбинации в необработанных клетках (ложных), клетках, обработанных миРНК отрицательного контроля (миОтр), миРНК против факторов НГСК (миРНК против Ku70, Ku80 и каталитической субъединицы ДНК-зависимой протеинкиназы (миKu70+80+ДНК-ПКкс)) и миРНК против ГР (миRad51). На дорожках GFP показан положительный контроль экспрессирующих GFP клеток. На дорожках, обозначенных «нерасщепленные ГР» и «нерасщепленные НГСК», показаны клетки отрицательного контроля, т.е. клетки, трансфицированные кольцевыми репортерными плазмидами ГР и НГСК. На дорожках, обозначенных «ГР I-Scel» и «НГСК I-Scel», представлены клетки, трансфицированные расщепленными Scel репортерными плазмидами, которые восстанавливают экспрессию GFP после репарации расщепления ДНК путем гомологичной рекомбинации или негомологичного соединения концов, соответственно. На данной фигуре показана эффективность ингибирования ГР или НГСК молекулами миРНК, о чем свидетельствует процент GFP-положительных клеток, который нормировали на процент dsRed-положительных клеток и выразили в виде кратности изменения относительно процента контрольных экспрессирующих GFP клеток, который приравняли к 1. Среднее значение для 3 экспериментов, усы показывают стандартную ошибку среднего. Статистическую значимость определяли с помощью непарного критерия Стьюдента; уровень значимости р<0,05 (*) и р<0,01 (**).
Фиг. 14. Влияние элементов MAR на подавление НГСК молекулами миРНК.
Показана кратность повышения экспрессии и встраивания GFP в клетки СНО, обработанные молекулами миРНК, направленными против факторов НГСК, и повторно трансфицированные плазмидами GFP или MAR-GFP. Средняя флуоресценция GFP, число копий и флуоресценция на копию GFP показаны в виде кратности превышения результата, полученного для необработанных клеток (обозначенных как «ложные»), трансфицированных плазмидой GFP. А) Результаты проточной цитометрии, В) анализ числа копий GFP в геноме с помощью количественной ПЦР, С) средняя флуоресценция каждого встроенного гена GFP (рассчитанная для каждого эксперимента как отношение экспрессии к числу копий). Среднее значение для 3 или более экспериментов; статистическую значимость определяли с помощью непарного критерия Стьюдента. Звездочками указаны значимые различия между обработанным миРНК образцом и соответствующим необработанным контролем; уровни значимости: p<0,05 (*), p<0,01 (**); усы показывают стандартную ошибку среднего.
Фиг. 15. Влияние элементов MAR на подавление ГР молекулами миРНК.
Показана кратность повышения экспрессии и встраивания GFP в клетки СНО, обработанные молекулами миРНК, направленными против факторов ГР, и повторно трансфицированные плазмидами GFP или MAR-GFP. Средняя флуоресценция GFP, число копий и флуоресценция на копию GFP показаны в виде кратности превышения результата, полученного для необработанных клеток (обозначенных как «ложные»), трансфицированных плазмидой GFP. А) Результаты проточной цитометрии, В) анализ числа копий GFP в геноме с помощью количественной ПЦР, С) средняя флуоресценция каждого встроенного гена GFP (рассчитанная для каждого эксперимента как отношение экспрессии к числу копий). Среднее значение для 3 или более экспериментов; статистическую значимость определяли с помощью непарного критерия Стьюдента. Звездочками указаны значимые различия между обработанным миРНК образцом и соответствующим необработанным контролем; уровни значимости: р<0,05 (*), р<0,01 (**); усы показывают стандартную ошибку среднего.
Фиг. 16. Влияние элементов MAR на подавление ОМСК молекулами миРНК.
Показана экспрессия и встраивание GFP в клетки СНО, обработанные молекулами миРНК, направленными против факторов ОМСК (и некоторых факторов ГР), и повторно трансфицированные плазмидами GFP (А) или MAR-GFP (В). Средняя флуоресценция GFP, число копий и флуоресценция на копию GFP показаны в виде кратности превышения результата, полученного для необработанных клеток (обозначенных как «ложные»), трансфицированных плазмидой GFP. На фигурах показаны результаты проточной цитометрии. Показано среднее значение для количества экспериментов, указанных внизу под графиком. Клетки, трансфицированные миMDC1, экспрессировали GFP даже без MAR на уровне, в 11,8 раз превышающем таковой для клеток, не трансфицированных миMDC. Особенно хороших результатов также можно было добиться для некоторых плазмид, которые не содержали MAR, а именно, для миBard1 и миLigl.
Фиг. 17. Влияние опосредованного миРНК подавления белка ГР.
На фигуре показано, что можно добиться более высокой экспрессии GFP и иммуноглобулина клетками СНО-М, стабильно экспрессирующими кшРНК, направленную против Rad51. Клетки СНО-М трансфицировали вектором экспрессии, полученным на основе транспозона PiggyBac, экспрессирующим кшРНК, направленную против Rad51, и полученную поликлональную популяцию клеток, а также клоны клеток, полученные из нее, повторно трансфицировали плазмидой, экспрессирующей GFP, наряду с исходными клетками СНО-М. Флуоресценцию GFP исходных СНО-М, популяции клеток, экспрессирующих Rad51-кшPHK, и полученных клонов оценивали через 10 дней после отбора на основании стабильной экспрессии гена GFP и гена устойчивости к пуромицину. Профили флуоресценции двух наиболее флуоресцирующих клонов показаны рядом с таковыми для объединенной популяции клеток и исходных клеток (А), а также процент клеток в секторах М1, М2 и М3 через 10 дней после отбора на основании устойчивости к пуромицину (В), который изображен на панели А в виде горизонтальных полос, отмеченных как 1, 2 и 3. Количественное соотношение клеток МЗ с высоким уровнем экспрессии отслеживали в течение дополнительных 68 дней культивирования без селекции, чтобы показать, что можно получить более стабильную экспрессию на более высоком уровне в клонах клеток, экспрессирующих кшРНК, по сравнению с исходными клетками СНО-М (С). В качестве альтернативы, экспрессионными плазмидами, кодирующими легкие и тяжелые цепи антитела инфликсимаба, трансфицировали типичные клоны, и оценивали удельную продуктивность секретируемого иммуноглобулина после селекции в течение дополнительных трех недель культивирования без антибиотика.
Фиг. 18. Влияние различных рекомбинантных элементов MAR человека, расположенных в 3'-5' направлении, на процентиль клеток с высоким и очень высоким уровнем продукции (% М3/М2), которое оценивали с помощью анализа методом сортировки клеток с возбуждением флуоресценции (FACS) по флуоресценции GFP для двух конструкций MAR.
(А) Указанные элементы MAR представляли собой перестроенные производные MAR Х-29 (X_29R2 (SEQ ID NO: 9), X_29R3 (SEQ ID NO: 10)), MAR 1-42 (1_42R2Bis, 1_42R3), MAR 1-6 (1_6R2 (SEQ ID NO: 8), 1_6R3) или MAR 1-68 (1_68R2 (SEQ ID NO: 7)), о чем свидетельствуют названия указанных конструкций. (В) Типичные профили FACS, полученные для лучших расположенных в 3'-5' направлении элементов MAR (MAR 1_68R (SEQ ID NO: 6)).
Фиг. 19. Стабильность экспрессии для двух векторов MAR.
Поликлональные популяции, сконструированные из векторов, содержащих производные MAR 1_68R2, 1_6R2 и X_29R3, исследовали в течение 5 недель культивирования без селекции, и еженедельно оценивали флуоресценцию GFP на протяжении данного периода. Оценивали процентиль субпопуляции МЗ: элемент MAR 1_6R2, расположенный в 3'-5' направлении, и неперестроенный MAR 1-68, расположенный в 5'-3' направлении, представляли собой лучшую исследованную комбинацию для вектора с двумя элементами MAR. Также показаны субпопуляции М1 и М2.
Фиг. 20. Векторы экспрессии, содержащие единичный генетический элемент.
Исследовали MAR 1_68 и Х_29 и использовали их в комбинации с репликатором LmnB2. Элементы MAR расположили в 5'-3' направлении от кассеты экспрессии трансгена, и оценивали в анализе трансфекции трансгеном на протяжении двух месяцев. Поликлональную популяцию стабильно трансфицированных клеток подвергали отбору по устойчивости к антибиотику в течение двух недель и исследовали флуоресценцию GFP с помощью анализа методом сортировки клеток с возбуждением флуоресценции (FACS) в течение семи недель. Соотношение клеток МЗ с высоким уровнем продукции показано на графике (А), тогда как на графике (В) показаны типичные профили FACS.
Фиг. 21. Векторы экспрессии, содержащие единичный генетический элемент Х-29.
Анализ стабильности вектора Х_29: показали, что вектор экспрессии, содержащий единичный Х_29 в 5'-3' направлении от кассеты экспрессии, оказался стабильным и позволял получить очень высокую процентиль субпопуляций М2 и М3 даже через 14 недель культивирования (27 пассажей).
Фиг. 22. Сравнительный анализ стабильно трансфицированных популяций СНО после 24 недель селекции антибиотиком.
Вектор с единичным MAR Х_29, расположенным в 5'-3' направлении от кассеты экспрессии (Puro_CGAPD_GFP_гастрин_X29), повышает появление клеток, экспрессирующих высокий уровень GFP, а также стабильность экспрессии с течением времени по сравнению с вектором с двумя элементами MAR: с 1_6R2 в качестве MAR, расположенного в 3'-5' направлении, и 1_68 в качестве MAR, расположенного в 5'-3' направлении (Puro_1_6R2_CGAPD_GFP_гастрин_1_68).
ПОДРОБНОЕ ОПИСАНИЕ РАЗЛИЧНЫХ И ПРЕДПОЧТИТЕЛЬНЫХ ВАРИАНТОВ
РЕАЛИЗАЦИИ НАСТОЯЩЕГО ИЗОБРЕТЕНИЯ
Термин трансген, используемый в контексте настоящего изобретения, представляет собой изолированную последовательность дезоксирибонуклеотидов (ДНК), кодирующую данный зрелый белок (также называемую в данном тексте ДНК, кодирующей белок), предшественника белка или функциональную РНК, которая не кодирует белок (некодирующую РНК). Трансген выделяют и встраивают в клетку для получения продукта трансгена. Некоторые предпочтительные трансгены согласно настоящему изобретению представляют собой трансгены, кодирующие иммуноглобулины (Ig), Fc-слитые белки и другие белки, в частности, белки с терапевтической активностью ("биотерапевтические средства"). Например, некоторые иммуноглобулины, такие как инфликсимаб (ремикейд) или другие секретируемые белки, такие как фактор коагуляции VIII, достаточно трудно экспрессировать вследствие клеточных затруднений, преимущественно неохарактеризованных. С помощью рекомбинантных молекул нуклеиновых кислот, векторов и способов согласно настоящему изобретению данные затруднения можно обнаружить и/или раскрыть. Это, как правило, позволяет повысить количество терапевтических белков, которые можно получить, и/или их качество, например, их процессинг и гомогенность посттрансляционных модификаций, таких как гликозилирование.
В данном тексте термин трансген не должен, в контексте ДНК, кодирующей белок, включать нетранскрибируемые, фланкирующие участки, такие как сигналы инициации транскрипции РНК, сайты полиаденилирования, промоторы или энхансеры. Другие предпочтительные трансгены включают последовательности ДНК, кодирующие функциональные РНК. Таким образом, термин трансген в контексте настоящего изобретения относится к последовательности ДНК, которую вводят в клетку, такую как эукариотическая клетка-хозяин, путем трансфекции (которая включает, в контексте настоящего изобретения, также трансдукцию, т.е., внедрение посредством вирусных векторов), и которая кодирует интересующий продукт, также называемый в данном тексте "продуктом экспрессии трансгена", например, "гетерологичными белками". Указанный трансген может быть функционально присоединен к последовательности, которая кодирует сигнальный пептид, который, в свою очередь, опосредует и/или способствует перемещению и/или секреции через эндоплазматический ретикулум и/или цитоплазматическую мембрану и удаляется до или в процессе секреции.
Малые интерферирующие РНК (миРНК) представляют собой двунитевые молекулы РНК, как правило, длиной 20-25 пар оснований, которые играют роль в РНК-интерференции (РНКи) путем интерференции с экспрессией определенных генов с комплементарной последовательностью нуклеотидов. миРНК можно непосредственно ввести в клетки или можно экспрессировать в клетке посредством вектора. Изолированная миРНК ТЕР, упоминаемая в данном тексте, представляет собой такую миРНК длиной 20-25 пар оснований, которую обычно вводят непосредственно в клетку, т.е., без экспрессии посредством нуклеиновой кислоты, которую внедрили в клетку.
Малая/короткая шпилечная РНК (кшРНК) представляет собой последовательность РНК, которая образует плотный шпилечный изгиб, который можно применять для замалчивания экспрессии целевого гена посредством РНКи. Экспрессию кшРНК в клетках обычно осуществляют путем доставки плазмид или вирусных векторов, таких как ретровирусные векторы. Для того, чтобы получить молекулы кшРНК, последовательность миРНК обычно модифицируют, чтобы ввести короткую петлю между двумя нитями миРНК. Нуклеиновую кислоту, кодирующую кшРНК, затем доставляют посредством вектора в клетку, и она транскрибируется в виде короткой шпилечной РНК (кшРНК), которую, возможно, дайсер обычным способом процессирует в функциональную миРНК.
Ми/кшРНК способна зависимым от последовательности способом снижать экспрессию целевого гена. КшРНК может гибридизоваться с участком транскрипта мРНК, кодирующего продукт целевого гена, тем самым ингибируя экспрессию целевого гена посредством РНК-интерференции. Бифункциональные кшРНК имеют больше одной мишени, например, кодирующую область, а также некоторые нетранслируемые области мРНК. Встраивание в геном клетки способствует длительному или конститутивному замалчиванию гена, которое может распространяться на дочерние клетки.
МикроРНК (миРНК) представляет собой малую молекулу РНК, например, длиной от 20 до 24 нуклеотидов, в частности, 22 нуклеотида, которая участвует в транскрипционной и посттранскрипционной регуляции экспрессии генов посредством спаривания с комплементарными последовательностями внутри мРНК. Замалчивание гена может происходить либо путем ингибирования транскрипции трансгена, либо путем деградации мРНК, либо путем предотвращения трансляции мРНК. МикроРНК можно экспрессировать путем доставки плазмид или вирусных векторов, таких как ретровирусные векторы. В качестве альтернативы, можно синтезировать молекулы РНК, ингибирующие или имитирующие микроРНК, и непосредственно трансфицировать ими клетки.
"Последовательность, кодирующая белок экспрессии/процессинга трансгена (ТЕР) или функциональную РНК ТЕР" позволяет экспрессировать или экспрессировать на повышенном уровне данный белок ТЕР после его переноса в клетку, тогда как последовательность, кодирующая некодирующие функциональные молекулы РНК, ингибирует экспрессию клеточных белков, соответственно. Белки ТЕР могут быть идентичны или аналогичны клеточным белкам, или они могут представлять собой белки из отличной клетки или вида. Клеточные белки, экспрессия которых, например, ингибируется функциональными молекулами РНК, представляют собой конститутивные белки клетки, в которую ввели функциональные молекулы РНК. Белок ТЕР также может дополнять экспрессию другого клеточного белка и, в результате, предпочтительно, повышает экспрессию трансгена. Указанные белки могут участвовать в рекомбинации; в процессах трансляции мРНК; в перемещении в ЭР, секреции, процессинге или фолдинге полипептидов, в транспорте ЭР-Гольджи-плазматическая мембрана, в гликозилировании и/или другой посттрансляционной модификации. Функциональные молекулы РНК включают, например, миРНК, кшРНК, микроРНК, сплайсированную в форме лассо РНК, малую временную смысловую РНК (мвРНК), антисмысловую РНК (аРНК), РНК рибозима и другие РНК, в частности, такие, которые могут подавлять экспрессию целевого гена. В особо предпочтительном варианте реализации данные белки вовлечены в "путь секреции белка" или в "пути рекомбинации", но также включают некоторые белки процессинга белка или метаболические белки, описанные ниже.
Функциональные РНК ТЕР можно не только экспрессировать с последовательности нуклеиновой кислоты, описанной выше, но их можно непосредственно ввести в клетку. Это, в частности, справедливо для изолированных миРНК ТЕР.
Термин "изолированная молекула нуклеиновой кислоты" в контексте настоящего изобретения эквивалентен термину "рекомбинантная молекула нуклеиновой кислоты", т.е. молекула нуклеиновой кислоты, которая не существует в данной форме в природе, но которую сконструировали из частей, которые существуют в природе.
Последовательность нуклеиновой кислоты, такая как ДНК или РНК, комплементарна другой ДНК или РНК, если нуклеотиды, например, из двух нитей однонитевых ДНК или двух нитей однонитевых РНК, могут образовывать стабильные водородные связи, например, водородную связь между гуанином (G) и цитозином (С). В клетке комплементарное спаривание оснований позволяет, например, клеткам копировать информацию от одного поколения к другому. При РНК-интерференции (РНКи) комплементарное спаривание оснований позволяет замалчивание или полный нокаут некоторых целевых генов. По существу, последовательность миРНК, кшРНК или микроРНК специфично снижает или нарушает экспрессию целевого гена путем выравнивания одинарной нити РНК (например, антисмысловой нити в миРНК) с РНК, в частности, мРНК клетки-хозяина. Степень комплементарности между двумя нитями нуклеиновой кислоты может изменяться от полной комплементарности (каждый нуклеотид напротив противоположного) до частичной комплементарности (50%, 60%, 70%, 80%, 90% или 95%). Степень комплементарности определяет стабильность комплекса и, таким образом, то, насколько успешно ген можно, например, нокаутировать. Таким образом, полная или по меньшей мере 95% комплементарность является предпочтительной.
Активность молекул миРНК при РНКи в значительной мере зависит от их способности связываться с индуцируемым РНК комплексом сайленсинга (RISC). За связыванием дуплекса миРНК с RISC следует разворачивание и расщепление смысловой нити эндонуклеазами. Оставшийся комплекс антисмысловая нить-RISC затем может связываться с целевыми мРНК для инициации замалчивания транскрипции.
В контексте настоящего изобретения трансгены, определение которых приведено выше, экспрессируют, как правило, белки, которые желательно получить в больших количествах, например, для применения в фармацевтике, тогда как последовательности, кодирующие белки ТЕР/функциональные РНК ТЕР, или сами функциональные молекулы РНК, разработаны таким образом, чтобы помогать экспрессии таких трансгенов либо непосредственно, либо опосредованно. "Типичный перечень белков ТЕР, экспрессированных с применением векторов, несущих транспозоны", приведен в таблице А. Для специалиста в данной области должно быть очевидно, что огромное количество таких белков было описано в данной области, и в таблице А представлены как идентификационные номера последовательностей NCBI для соответствующих белков, так и последовательности нуклеиновых кислот, кодирующие их. В последней колонке приведены идентификаторы последовательностей для некоторых из данных последовательностей. Для специалиста в данной области должно быть очевидно, что варианты белков, а также последовательности, идентичные им более чем на 80%, 90%, 95% или 98%, входят в объем настоящего изобретения.
"Типичный перечень кшРНК, экспрессируемых с применением, например, определенных векторов, несущих транспозоны piggybac", приведен в таблице В. Для специалиста в данной области должно быть очевидно, что такие кшРНК можно легко сконструировать после того, как выбран целевой ген. Например, любой из известных генов пути рекомбинации представляет собой готовый целевой ген. Тем не менее, другие гены, такие как гены, кодирующие белки, представленные в таблице А, могут представлять собой готовые мишени для миРНК, полученных из таких кшРНК. Таблица С представляет собой перечень примеров миРНК (смысловая нить) и примеров кшРНК, полученных из соответствующих миРНК. Антисмысловую нить миРНК в конечном счете применяют, чтобы блокировать и/или вызвать деградацию клеточной мРНК. Это, как правило, приводит к снижению уровней белка, кодируемого указанной мРНК.
Идентичность означает степень родства между двумя последовательностями нуклеотидов, которую определяют по идентичности пар между двумя нитями таких последовательностей, например, целой и полной последовательности. Идентичность можно легко рассчитать. Несмотря на то, что существует множество способов измерения идентичности между двумя последовательностями нуклеотидов, термин "идентичность" хорошо известен квалифицированным специалистам (Computational Molecular Biology, Lesk, A.M., ред., Oxford University Press, Нью-Йорк, 1988; Biocomputing: Informatics and Genome Projects, Smith, D.W., ред., Academic Press, Нью-Йорк, 1993; Computer Analysis of Sequence Data, часть I, Griffin, A.M., и Griffin, H.G., ред., Human Press, Нью-Джерси, 1994; Sequence Analysis in Molecular Biology, von Heinje, G., Academic Press, 1987; и Sequence Analysis Primer, Gribskov, M. и Devereux, J., ред., M Stockton Press, Нью-Йорк, 1991). Способы, широко используемые для определения идентичности между двумя последовательностями, включают, но не ограничены способами, описанными в Guide to Huge Computers, Martin J. Bishop, ред., Academic Press, Сан-Диего, 1994, и Carillo, H., и Lipman, D., SIAM J Applied Math. 48: 1073 (1988). Разработаны предпочтительные способы определения идентичности, чтобы получить наибольшие совпадения между двумя исследуемыми последовательностями. Такие способы закодированы в компьютерных программах. Предпочтительные способы на основе компьютерных программ для определения идентичности между двумя последовательностями включают, но не ограничены пакетом программ GCG (Genetics Computer Group, Мэдисон, Висконсин) (Devereux, J., и др., Nucleic Acids Research 12(1). 387 (1984)), BLASTP, BLASTN, FASTA (Altschul и др. (1990); Altschul и др. (1997)). Хорошо известный алгоритм Smith Waterman также можно применять для определения идентичности.
В качестве примера, нуклеиновая кислота, включающая последовательность нуклеотидов, "идентичную" по меньшей мере, например, на 95% эталонной последовательности нуклеотидов, означает, что последовательность нуклеотидов нуклеиновой кислоты идентична эталонной последовательности за исключением того, что указанная последовательность нуклеотидов может содержать до пяти точечных мутаций на каждые 100 нуклеотидов эталонной последовательности нуклеотидов. Другими словами, чтобы получить последовательность нуклеотидов, по меньшей мере на 95% идентичную эталонной последовательности нуклеотидов, до 5% нуклеотидов в указанной эталонной последовательности можно удалить или заменить на другой нуклеотид, или множество нуклеотидов, составляющее до 5% от всех нуклеотидов в эталонной последовательности, можно вставить в эталонную последовательность. Данные мутации эталонной последовательности могут встречаться в 5'- или 3'-концевых положениях эталонной последовательности нуклеотидов или в любом месте между этими концевыми положениями, либо разбросанные отдельно среди нуклеотидов в эталонной последовательности, либо расположенные в одной или более непрерывных группах внутри эталонной последовательности. Идентичности последовательностей более чем на приблизительно 60%, приблизительно 70%, приблизительно 75%, приблизительно 85% или приблизительно 90% для любой последовательности, описанной в данной заявке (например, путем указания SEQ ID и/или номера доступа), также входят в объем настоящего изобретения.
Последовательность нуклеиновой кислоты, по существу идентичная другой последовательности нуклеиновой кислоты, относится к последовательности, содержащей точечные мутации, делеции или вставки в последовательности, которые не оказывают или оказывают несущественное влияние на соответствующий описанный способ и часто представлены одной, двумя, тремя или четырьмя мутациями на 100 п.о.
Настоящее изобретение относится к вариантам как полинуклеотида, так и полипептида. "Вариант" относится к полинуклеотиду или полипептиду, отличающемуся от описанного полинуклеотида или полипептида, но сохранившему его существенные свойства. Как правило, варианты в целом очень сходны и на многих участках идентичны полинуклеотиду или полипептиду согласно настоящему изобретению. Указанные варианты могут включать изменения в кодирующих областях, некодирующих областях или в обоих указанных типах областей. Особенно предпочтительны полинуклеотидные варианты, включающие изменения, которые вызывают молчащие замены, вставки или делеции, но не изменяют свойства или активности кодированного полипептида. Нуклеотидные варианты, полученные путем молчащих замен, вследствие вырожденности генетического кода, являются предпочтительными. Более того, варианты, в которых 5-10, 1-5 или 1-2 аминокислоты, описанные в данной заявке, заменены, удалены или вставлены в любой комбинации, также предпочтительны. В объем настоящего изобретения также входят аллельные варианты указанных полинуклеотидов. Аллельный вариант обозначает любую из двух или более альтернативных форм гена, занимающих один и тот же хромосомный локус. Аллельная вариация в природе возникает в результате мутации и может приводить к полиморфизму внутри популяции. Мутации в гене могут быть молчащими (без изменений в кодируемом полипептиде) или могут кодировать полипептиды, последовательности аминокислот которых изменены. Аллельный вариант полипептида представляет собой полипептид, кодируемый аллельным вариантом гена. Варианты любых из молекул нуклеиновых кислот, описанных в данной заявке, входят в объем настоящего изобретения.
Последовательность промотора или сам промотор представляет собой последовательность нуклеиновой кислоты, которая узнается клеткой-хозяином для экспрессии определенной последовательности нуклеиновой кислоты. Последовательность промотора включает последовательности, контролирующие транскрипцию, которые регулируют экспрессию указанного полинуклеотида. Промотор может представлять собой любую последовательность нуклеиновой кислоты, которая проявляет транскрипционную активность в выбранной клетке хозяина, включая мутантные, укороченные и гибридные промоторы, и его можно получить из генов, кодирующих внеклеточные или внутриклеточные полипептиды, либо гомологичные, либо гетерологичные для клетки хозяина. Промоторы согласно настоящему изобретению включают индуцируемые и неиндуцируемые промоторы. Последовательность нуклеиновой кислоты находится под контролем промотора, когда промотор выполняет свою функцию на указанной нуклеиновой кислоте.
CGAPDH (также называемый в данном тексте C_GAPDH) представляет собой слитые энхансер и промотор, которые включают промотор GAPDH человека и энхансер немедленно-раннего гена CMV человека. В одном варианте реализации для его получения промотор GAPDH человека и его 5'-нетранслируемую область (НТО) амплифицировали путем ПЦР из геномной ДНК клеток HEK293 человека. Полученный продукт помещали в 5'-3' направлении от энхансера немедленно-раннего гена CMV человека. См. пример последовательности в SEQ ID NO: 11. Последовательности, которые на по меньшей мере 80%, 90%, 95%, 98%, 99% или 100% идентичны последовательности SEQ ID NO: 11, также входят в объем настоящего изобретения. Другой желательный промотор(ы) и/или энхансер(ы) или продукты их слияния представляют собой, но не ограничены перечисленными: энхансер немедленно-раннего гена CMV человека, промотор GAPDH человека, промотор EF1-альфа человека, промотор CMV, промотор SV40, промотор Actb СНО или промотор Hspa5 СНО. Данные элементы хорошо известны в данной области, и примеры последовательностей перечислены в SEQ ID NO: 110-116. Для специалиста в данной области должно быть очевидно, что их варианты также входят в объем настоящего изобретения, также, как и элемент, последовательность которого по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентична любой из последовательностей SEQ ID NO: 110-116.
"Транспозон" представляет собой мобильный генетический элемент, который эффективно перемещается между векторами и хромосомами с помощью механизма "вырезать и вставить" или "копировать и вставить". В процессе транспозиции транспозаза (например, транспозаза РВ в системе транспозона PiggyBac) узнает транспозон-специфичные последовательности инвертированных концевых повторов (ITR), расположенные на обоих концах транспозона (существуют 5'- и а 3'-ITR в любой системе транспозона), перемещает содержимое из исходных сайтов и встраивает их в хромосомные сайты, такие как хромосомные сайты ТТАА. Мощная активность системы транспозона PiggyBac позволяет легко перемещать интересующие гены, расположенные между двумя ITR, в целевые геномы. Система транспозона PiggyBac описана, например, в 2010/0154070, которая полностью включена в данную заявку посредством ссылки.
Элементы MAR (конструкции MAR, последовательности MAR, S/MAR или просто MAR) принадлежат к более широкой группе эпигенетических регуляторных элементов, которые также включают граничные или инсуляторные элементы, такие как cHS4, регуляторные области локусов (LCR), стабилизирующие и антирепрессорные (STAR) элементы, универсальные элементы, открывающие хроматин (UCOE), или модификаторы гистонов, такие как гистоновая дезацетилаза (HDAC).
Элементы MAR могут быть определены на основании идентифицированного MAR, на котором они первично основаны: конструкция MAR S4, соответственно, представляет собой элементы MAR, большинство нуклеотидов которых (50% и более, предпочтительно 60%, 70% или 80%) основаны на MAR S4. Некоторые простые мотивы последовательности, такие как мотивы с высоким содержанием А и Т, часто обнаруживают внутри элементов MAR. Другие широко распространенные мотивы представляют собой мотивы А-бокс, Т-бокс, мотивы раскручивания спирали ДНК, сайты связывания SATB1 (Н-бокс, A/T/С25) и консенсусные сайты топоизомеразы II для позвоночных или дрозофилы.
MAR, как правило, описывают как последовательности в ДНК эукариотических хромосом, к которым присоединяется ядерный матрикс. Свойства MAR лишь отчасти определяются их первичной структурой. Например, известно, что типичная первичная структура, обнаруженная в элементах MAR, таких как АТ-богатые области, приводит к образованию третичных структур, а именно, некоторых изгибов, которые определяют функцию MAR. Таким образом, MAR часто определяют не только по их первичной структуре, но также по их вторичной, третичной структуре, например, степени их изогнутости и/или физическим свойствам, таким как температура плавления.
Обогащенная АТ/TA-динуклеотидами область изгиба ДНК (здесь и далее называемая "AT-богатой областью"), обнаруживаемая в большинстве элементов MAR, представляет собой область изгиба ДНК, содержащую большое количество А и Т, в частности, в виде динуклеотидов AT и ТА. В предпочтительном варианте реализации она содержит по меньшей мере 10% динуклеотида ТА, и/или по меньшей мере 12% динуклеотида AT на участке из 100 непрерывных пар оснований, предпочтительно, по меньшей мере 33% динуклеотида ТА, и/или по меньшей мере 33% динуклеотида AT на участке из 100 непрерывных пар оснований (или на соответствующем более коротком участке при меньшей длине АТ-богатой области), и при этом имеет изогнутую вторичную структуру. Тем не менее, "АТ-богатые области" могут быть настолько короткими, как приблизительно 30 нуклеотидов или менее, но предпочтительно их длина составляет приблизительно 50 нуклеотидов, приблизительно 75 нуклеотидов, приблизительно 100 нуклеотидов, приблизительно 150, приблизительно 200, приблизительно 250, приблизительно 300, приблизительно 350 или приблизительно 400 нуклеотидов или более.
Некоторые сайты связывания также часто имеют относительно высокое содержание А и Т, такие как сайты связывания SATB1 (Н-бокс, А/Т/С25) и консенсусные сайты топоизомеразы II для позвоночных (RNYNNCNNGYNGKTNYNY) или дрозофилы (GTNWAYATTNATNNR). Тем не менее, область сайта связывания (модуль), в частности, область TFBS, которая содержит кластер сайтов связывания, можно легко отличить от областей, обогащенных динуклеотидами AT и ТА ("АТ-богатых областей"), от элементов MAR, богатых содержанием А и Т, путем сравнения паттерна кривизны указанных областей. Например, средняя степень кривизны MAR 1_68 человека может превышать приблизительно 3,8 или приблизительно 4,0, тогда как средняя степень кривизны участка TFBS может быть ниже приблизительно 3,5 или приблизительно 3,3. Области идентифицированного MAR также можно подтвердить с помощью альтернативных способов, таких как, но не ограничиваясь определением относительных температур плавления, описанных в других местах в данной заявке. Тем не менее, такие значения видоспецифичны и, следовательно, могут изменяться от вида к виду и могут, например, быть ниже. Таким образом, градусы кривизны соответствующих областей, богатых динуклеотидами AT и ТА, могут быть ниже, например, от приблизительно 3,2 до приблизительно 3,4, или от приблизительно 3,4 до приблизительно 3,6, или от приблизительно 3,6 до приблизительно 3,8, и градусы кривизны областей TFBS могут быть пропорционально ниже, например, ниже приблизительно 2,7, ниже приблизительно 2,9, ниже приблизительно 3,1, ниже приблизительно 3,3. Квалифицированный специалист выберет, соответственно, меньшие размеры окна в программе SMAR Scan II.
Элемент MAR, конструкция MAR, последовательность MAR, S/MAR или просто MAR согласно настоящему изобретению представляет собой последовательность нуклеотидов, одно или более (например, два, три или четыре) свойств которой, таких как описанные выше, аналогичны таковым у встречающихся в природе "SAR" или "MAR". Предпочтительно у такого элемента MAR, конструкции MAR, последовательности MAR, S/MAR или просто MAR по меньшей мере одно свойство способствует экспрессии белка любого гена, находящегося под влиянием указанного MAR. Элемент MAR, как правило, также обладает свойством изолированной и/или очищенной нуклеиновой кислоты, предпочтительно проявляющей активность MAR, в частности, проявляющей активность модуляции транскрипции, предпочтительно активность усиления транскрипции, но также проявляющей, например, активность стабилизации экспрессии и/или другие активности.
Термины элемент MAR, конструкция MAR, последовательность MAR, S/MAR или просто MAR также включают, в некоторых вариантах реализации, улучшенные конструкции MAR, которые обладают свойствами, которые обеспечивают улучшение по сравнению со встречающимся в природе и/или идентифицированным MAR, на котором может быть основана конструкция MAR согласно настоящему изобретению. Такие свойства включают, но не ограничены перечисленными: уменьшенную длину по сравнению с полноразмерным встречающимся в природе и/или обнаруженным MAR, повышенную экспрессию гена/транскрипции, повышенную стабильность экспрессии, тканеспецифичность, способность к индуцированию или комбинацию перечисленных свойств. Соответственно, элемент MAR, который подвергли улучшению, например, содержит менее чем приблизительно 90%, предпочтительно менее чем приблизительно 80%, еще более предпочтительно менее чем приблизительно 70%, менее чем приблизительно 60%, или менее чем приблизительно 50% от количества нуклеотидов идентифицированной последовательности MAR. Элемент MAR может повышать экспрессию гена и/или транскрипцию трансгена после трансформации подходящей клетки указанной конструкцией.
Элемент MAR предпочтительно встраивают в 3'-5' направлении от промоторной области, с которой функционально связан или может быть функционально связан интересующий ген. Тем не менее, в некоторых вариантах реализации предпочтительно, чтобы элемент MAR был расположен как в 3'-5' направлении, так и в 5'-3' направлении, или только в 5'-3' направлении от интересующего гена/последовательности нуклеиновой кислоты. Другие разнообразные цис- и/или транс-расположения MAR также входят в объем настоящего изобретения.
Термин «синтетический», когда его применяют в контексте элемента MAR, относится к MAR, разработка которого включала больше изменений, чем простая перестановка, дупликация и/или делеция последовательностей/областей или части областей идентифицированных MAR или MAR, основанных на них. В частности, синтетические MAR/элементы MAR как правило включают одну или более, предпочтительно одну область идентифицированного MAR, которая, тем не менее, в некотором варианте реализации могла быть синтезирована или модифицирована, а также специально разработанные, хорошо охарактеризованные элементы, такие как единичный TFBS или серия TFBS, которые, в предпочтительном варианте реализации, получают синтетическим путем. Данные разработанные элементы во многих вариантах реализации относительно коротки, в частности, их длина, как правило, составляет не больше чем приблизительно 300 п.о., предпочтительно не больше чем приблизительно 100, приблизительно 50, приблизительно 40, приблизительно 30, приблизительно 20 или приблизительно 10 п.о. Данные элементы в некоторых вариантах реализации могут быть мультимеризованы. Такие синтетические элементы MAR также входят в объем настоящего изобретения, и должно быть очевидно, что, как правило, в настоящем описании следует понимать, что все, что указано как применимое к "элементу MAR", в равной степени применимо к синтетическому элементу MAR.
Функциональные фрагменты последовательностей нуклеотидов обнаруженных элементов MAR также включены при условии, что они сохраняют функции элемента MAR, описанные выше.
Некоторые предпочтительные идентифицированные элементы MAR включают, но не ограничены перечисленными: MAR 1_68, MAR Х_29, MAR 1_6, MAR S4, MAR S46, включая все их перестановки, описанные в WO 2005040377 и публикации патента США 20070178469, которые особенно включены в настоящую заявку посредством ссылки для описания последовательностей данных и других элементов MAR. MAR лизоцима цыпленка также представляет собой предпочтительный вариант реализации (см., патент США номер 7129062, который также особенно включен в данную заявку для описания элементов MAR).
Если указано, что вектор содержит единичный MAR, это означает, что внутри указанного вектора присутствует один MAR и нет других MAR либо такого же, либо отличного типа или структуры.
В некоторых вариантах реализации настоящего изобретения присутствует несколько MAR, которые могут иметь одинаковый или отличный тип или структуру, все из которых могут быть расположены в 5'-3' направлении от интересующего гена. Это называется кластером единичных MAR.
Если указано, что что-либо, например, количество клеток, стабильно экспрессирующих полипептид, "не зависит" от присутствия, например, последовательности, то указанная последовательность не оказывает влияния (например, на количество клеток, стабильно экспрессирующих полипептид) до любой статистически значимой степени.
Трансген или последовательность, кодирующая белок экспрессии/процессинга трансгена или функциональную РНК согласно настоящему изобретению, часто является частью вектора.
Вектор согласно настоящему изобретению представляет собой молекулу нуклеиновой кислоты, способную переносить другую нуклеиновую кислоту, такую как трансген, которую нужно экспрессировать с помощью данного вектора, с которым она была связана, как правило, в который она была встроена. Например, плазмида представляет собой тип вектора, ретровирус или лентивирус представляет собой другой тип вектора. В предпочтительном варианте реализации настоящего изобретения вектор линеаризуют перед трансфекцией. Вектор экспрессии содержит регуляторные элементы или находится под контролем таких регуляторных элементов, которые разработаны таким образом, чтобы способствовать транскрипции и/или экспрессии последовательности нуклеиновой кислоты, которую несет указанный вектор экспрессии. Регуляторные элементы включают энхансеры и/или промоторы, но также различные другие элементы, описанные в данной заявке (см. также раздел "Разработка вектора").
Последовательность вектора представляет собой последовательность ДНК или РНК указанного вектора, за исключением любых "других" нуклеиновых кислот, таких как трансгены, а также генетических элементов, таких как элементы MAR.
Эукариотическую клетку, включая клетку млекопитающего, такую как рекомбинантная клетка млекопитающего/эукариотическая клетка-хозяин, согласно настоящему изобретению можно поддерживать в условиях культуры клеток. Лишь некоторые из примеров данного типа клеток представляют собой эукариотические клетки-хозяева, не относящиеся к приматам, такие как клетки яичника китайского хомячка (СНО) и клетки почки детеныша хомячка (ВНК, АТСС CCL 10). Эукариотические клетки-хозяева примата включают, например, клетки карциномы шейки матки человека (HELA, АТСС CCL 2) и линию клеток почки обезьяны CV1, трансформированную SV40 (COS-7, АТСС CRL-1587). Рекомбинантная эукариотическая клетка-хозяин или рекомбинантная клетка млекопитающего означает клетку, которую модифицировали, например, путем трансфекции, например, трансгенной последовательностью, и/или путем мутации. Эукариотические клетки-хозяева или рекомбинантные клетки млекопитающих способны осуществлять посттранскрипционные модификации белков, экспрессируемых указанными клетками. В некоторых вариантах реализации настоящего изобретения клеточный аналог в эукариотической (например, не относящейся к примату) клетке-хозяине полностью функционален, т.е., не был, например, инактивирован путем мутации. Предпочтительно, чтобы трансгенная последовательность (например, примата) экспрессировалась дополнительно к ее клеточному аналогу (например, не относящемуся к примату).
Трансфекция согласно настоящему изобретению представляет собой внедрение нуклеиновой кислоты в реципиентную эукариотическую клетку, например, не ограничиваясь перечисленными, путем электропорации, липофекции, как правило с помощью невирусного вектора (опосредованная вектором трансфекция) или с помощью химических способов, включая применение поликатионных липидов. Трансфекция, не опосредованная вектором, включает, например, непосредственное внедрение изолированных миРНК ТЕР в клетку. Во временно трансфицированной клетке, например, миРНК сохраняется лишь временно. В контексте настоящего изобретения можно осуществить первую трансфекцию по меньшей мере одной молекулой нуклеиновой кислоты с последовательностью, кодирующей белок экспрессии/процессинга трансгена (ТЕР) или функциональную РНК ТЕР, или, в качестве альтернативы, непосредственно функциональной РНК ТЕР (например, миРНК), и вторую последующую трансфекцию нуклеиновой кислотой, кодирующей трансген. Как первую, так и вторую трансфекцию можно повторить. Например, миРНК, внедренная в процессе первой трансфекции, действует, в частности, ингибирует, рекомбинационный белок (белок, который участвует в событиях рекомбинации в трансфицированной клетке). После этого в процессе второй последующей трансфекции вводят трансген.
Транскрипция означает синтез РНК по матрице ДНК. 'Транскрипционно активный" относится, например, к трансгену, который транскрибируется. Трансляция представляет собой процесс, с помощью которого по РНК синтезируется белок.
Улучшение секреции измеряют по сравнению со значением, полученным для контрольной клетки, которая не содержит соответствующую трансгенную последовательность. Любое статистически значимое улучшение по сравнению со значением для контроля называют стимуляцией.
Маркер отбора представляет собой нуклеиновую кислоту, которая содержит ген, продукт которого придает устойчивость к селекционному агенту - антибиотику (например, хлорамфениколу, ампициллину, гентамицину, стрептомицину, тетрациклину, канамицину, неомицину, пуромицину) или способность расти на селективных средах (например, с DHFR (дигидрофолатредуктазой)).
Класс белков, известных как шапероны, определяют как белки, которые связываются и стабилизируют в противном случае нестабильный конформационный изомер другого белка и, путем контролируемого связывания и высвобождения, способствуют его правильной судьбе in vivo, будь то фолдинг, сборка олигомера, транспорт в конкретный клеточный компартмент или утилизация путем деградации. BiP (также известный как GRP78, белок, связывающий тяжелую цепь lg, и Kar2p в дрожжах) представляет собой распространенный шаперон массой приблизительно 70 кДа из семейства hsp 70, находящийся в эндоплазматическом ретикулуме (ER), который, среди прочих функций, способствует транспорту в секреторной системе и способствует фолдингу белков. Протеиндисульфидизомераза (PDI) представляет собой белок-шаперон, находящийся в ЭР, который участвует в катализе образования дисульфидной связи при посттрансляционном процессинге белков.
КЛЕТОЧНАЯ МЕТАБОЛИЧЕСКАЯ ИНЖЕНЕРИЯ
В клеточной метаболической инженерии, например, модифицируют процессы, свойственные экспрессирующей клетке. Например, некоторые белки секреторного пути, например, экспрессируют сверх меры. В качестве альтернативы, изменяют события рекомбинации путем влияния на пути рекомбинации.
ПУТЬ СЕКРЕЦИИ БЕЛКА
Секреция белков представляет собой процесс, общий для организмов из всех трех царств. Для такого сложного пути секреции в первую очередь требуется перемещение белка из цитозоля через цитоплазматическую мембрану клетки. Для достижения белком конечного назначения необходимо несколько этапов и различных факторов. В клетках млекопитающих такой путь секреции включает два основных макромолекулярных ансамбля: сигналузнающую частицу (SRP) и секреторный комплекс (Sec-комплекс или транслокон). SRP состоит из шести белков с массами 9, 14, 19, 54, 68 и 72 кДа и 7S РНК, а транслокон представляет собой тороидальную частицу, состоящую из Sec61αβy, Sec62 и Sec63. Номера доступа (в скобках), представленные для человеческого варианта некоторых из данных белков, описаны далее: hSRP14 (номер доступа Х73459.1); hSRP9 (NM_001130440); hSRP54 (NM_003136); hSRPRα (NM_003139); hSRPRβ (NM_021203); hSEC61α1 (NM_013336); hSEC61 β (L25085.1); hSEC61 γ (AK311845,1).
Первый этап секреции белка зависит от сигнальных пептидов, которые содержат определенную пептидную последовательность на аминоконце полипептида, которая опосредует перемещение образующегося белка через мембрану и в просвет эндоплазматического ретикулума (ЭР). На данном этапе сигнальный пептид, который выходит из лидирующей транслирующей рибосомы, взаимодействует с субъединицей частицы SRP, которая узнает сигнальный пептид, а именно, с SRP54. Связывание SRP с сигнальным пептидом блокирует дальнейшую элонгацию образующегося полипептида, что приводит к остановке трансляции. Белки SRP-9 и -14 необходимы для остановки элонгации (Walter и Blobel 1981). На втором этапе комплекс рибосома-образующийся полипептид-SRP стыкуется с мембраной ЭР посредством взаимодействия SRP54 с рецептором SRP (SR) (Gilmore, Blobel и др. 1982; Pool, Stumm и др. 2002). SR представляет собой гетеродимерный комплекс, содержащий два белка, SRα и SRβ, которые проявляют ГТФазную активность (Gilmore, Walter и др. 1982). Взаимодействие SR с SRP54 зависит от связывания ГТФ (Connolly, Rapiejko и др. 1991). SR координирует высвобождение SRP из комплекса рибосома-образующийся полипептид и ассоциацию сайта выхода из рибосомы с комплексом Sec61 (транслоконом). Растущий образующийся полипептид проникает в ЭР через канал транслокона, и трансляция возобновляется с обычной скоростью. Рибосома остается связанной на цитоплазматической стороне транслокона до тех пор, пока трансляция не завершится. Дополнительно к рибосомам, транслоконы тесно связаны с рибофорином на цитоплазматической стороне и с шаперонами, такими как кальретикулин и кальнексин, и протеиндисульфидизомеразами (PDI) и олигосахарид-трансферазой на стороне просвета ЭР. После внедрения растущего образующегося полипептида в просвет ЭР сигнальный пептид отщепляется от пре-белка ферментом, называемым сигнальной пептидазой, что приводит к высвобождению зрелого белка в ЭР. После посттрансляционной модификации, правильному фолдингу и мультимеризации белки покидают ЭР и мигрируют в аппарат Гольджи, а затем в секреторные везикулы. Слияние секреторных везикул с плазматической мембраной приводит к высвобождению содержимого везикулы во внеклеточное пространство.
Удивительно, что секретируемые белки в ходе эволюции обрели определенные сигнальные последовательности, которые хорошо подходят для их перемещения через мембрану клетки. Различные последовательности, обнаруженные как отдельные сигнальные пептиды, могут взаимодействовать уникальными путями с секреторным аппаратом. Сигнальные последовательности преимущественно гидрофобны по своей природе, и это свойство может способствовать получению из образующегося пептида секреторного белка. Дополнительно к гидрофобному участку аминокислот последовательности большинства сигналов секреции млекопитающих обладают множеством общих свойств. Различные сигнальные пептиды отличаются по эффективности, с которой они направляют секрецию гетерологичных белков, но были обнаружены несколько сигнальных пептидов секреции (т.е. сигнальных последовательностей из интерлейкина, иммуноглобулина, рецептора гистосовместимости и т.д.), которые можно применять, чтобы направлять секрецию гетерологичных рекомбинантных белков. Несмотря на сходства, данные последовательности не оптимальны для стимуляции эффективной секреции некоторых белков, которые трудно поддаются экспрессии, вследствие того, что нативный сигнальный пептид не может правильно функционировать вне нативного контекста, или из-за различий, связанных с клеткой-хозяином или процессом секреции. Выбор подходящей сигнальной последовательности для эффективной секреции гетерологичного белка может дополнительно осложняться взаимодействием последовательностей внутри отщепленного сигнального пептида с другими участками зрелого белка (Johansson, Nilsson и др. 1993).
ПУТИ РЕКОМБИНАЦИИ
Пути рекомбинации, также известные как пути рекомбинации ДНК, представляют собой клеточные пути, которые приводят к репарации повреждения ДНК, например, к соединению концов молекулы ДНК после двухнитевых разрывов хромосомы и к обмену или слиянию последовательностей ДНК между хромосомными и нехромосомными молекулами ДНК, как, например, при кроссинговере хромосом при мейозе или перестройке генов иммуноглобулинов в лимфоцитарных клетках. Существует три основных пути рекомбинации: путь гомологичной рекомбинации (ГР), путь негомологичного соединения концов (НГСК), путь опосредованного микрогомологией соединения концов (ОМСК) и альтернативного соединения концов (Альт-СК).
МЕХАНИЗМЫ ГОМОЛОГИЧНОЙ РЕКОМБИНАЦИИ (ГР), НЕГОМОЛОГИЧНОГО СОЕДИНЕНИЯ КОНЦОВ (НГСК) И ОПОСРЕДОВАННОГО МИКРОГОМОЛОГИЕЙ СОЕДИНЕНИЯ КОНЦОВ (ОМСК)
Трансгены используют аппарат рекомбинации для встраивания в двухнитевой разрыв в геноме хозяина.
Двухнитевые разрывы (ДНР) представляют собой биологически наиболее разрушительный тип геномного повреждения, потенциально приводящий к тбели клетки или большому разнообразию генетических перестроек. Точная репарация необходима для успешного поддержания и передачи генетической информации.
Существует два основных механизма репарации ДНР: негомологичное соединение концов (НГСК) и гомологичная рекомбинация (ГР). Третий механизм, называемый опосредованным микрогомологией соединением концов (ОМСК), часто начинает действовать, когда два основных механизма репарации ДНР претерпевают неудачу. Гомологичная рекомбинация представляет собой процесс генетического обмена между последовательностями ДНК, которые гомологичны друг другу, и действует преимущественно в течение фаз S/G2 клеточного цикла, тогда как НГСК просто соединяет два разорванных конца ДНК, обычно при отсутствии гомологии последовательностей, и действует на всех фазах клеточного цикла, но имеет особое значение во время G0-G1 и ранней S фазы митотических клеток (Wong и Capecchi, 1985; Delacote и Lopez, 2008). У позвоночных ГР, НГСК и ОМСК различным образом участвуют в репарации ДНР, в зависимости от природы ДНР и фазы клеточного цикла (Takata и др., 1998).
НГСК: основные механизмы
По существу, молекулярный механизм процесса НГСК кажется простым: 1) набор ферментов захватывает разорванную молекулу ДНК, 2) образуется молекулярный мостик, который сближает два конца ДНК, и 3) разорванные молекулы заново лигируются. Аппарат НГСК в клетках млекопитающих включает два белковых комплекса для осуществления таких реакций: гетеродимер Ku80/Ku70, связанный с ДНК-ПКкс (каталитической субъединицей ДНК-зависимой протеинкиназы), и ДНК-лигаза IV с ее кофактором XRCC4 (комплементирующий ген 4 репарации рентгеновских повреждений у китайского хомячка) и множеством белковых факторов, таких как Artemis (Артемида) и XLF (XRCC4-подобный фактор; или Cernunnos (Цернун)) ( и др., 2002). НГСК часто считают допускающая ошибки репарацией ДНР, так как она просто соединяет два разорванных конца ДНК, обычно при отсутствии гомологии последовательностей, и порождает небольшие вставки и делеции (Moore и Haber, 1996; Wilson и др., 1999). НГСК обеспечивает механизм репарации ДНР на всем протяжении клеточного цикла, но особое значение имеет в течение G0-G1 и ранней S-фазы митотических клеток (Takata и др., 1998; Delacote и Lopez, 2008). Репарацию ДНР посредством НГСК наблюдают в организмах начиная с бактерий и заканчивая млекопитающими, что указывает на ее консервативный характер в процессе эволюции.
и др., 2002). НГСК часто считают допускающая ошибки репарацией ДНР, так как она просто соединяет два разорванных конца ДНК, обычно при отсутствии гомологии последовательностей, и порождает небольшие вставки и делеции (Moore и Haber, 1996; Wilson и др., 1999). НГСК обеспечивает механизм репарации ДНР на всем протяжении клеточного цикла, но особое значение имеет в течение G0-G1 и ранней S-фазы митотических клеток (Takata и др., 1998; Delacote и Lopez, 2008). Репарацию ДНР посредством НГСК наблюдают в организмах начиная с бактерий и заканчивая млекопитающими, что указывает на ее консервативный характер в процессе эволюции.
После образования ДНР ключевым этапом в пути репарации НГСК является физическое сближение разорванных концов ДНК. НГСК запускается при взаимодействии гетеродимерного белкового комплекса Ku70/80 с обоими концами разорванной молекулы ДНК, чтобы захватить, сблизить концы и создать каркас для сборки других ключевых факторов НГСК. Связанный с ДНК гетеродимерный комплекс Ku привлекает в место ДНР ДНК-ПКкс, белок массой 460 кДа, принадлежащий к семейству PIKK (фосфоинозитид-3-киназа-подобному семейству протеинкиназ) (Gottlieb и Jackson, 1993), и активирует его серин/треонинкиназную функцию (Yaneva и др., 1997). Две молекулы ДНК-ПКкс с двух концов ДНР взаимодействуют, таким образом образуя молекулярный мостик между обоими разорванными концами ДНК и ингибируя их деградацию (DeFazio и др., 2002). Затем концы ДНК можно непосредственно лигировать, хотя большинство концов, возникших в результате ДНР, должны быть надлежащим образом процессированы перед лигированием (Nikjoo и др., 1998). В зависимости от природы разрыва может потребоваться действие различных комбинаций ферментов процессинга для получения совместимых выступов путем заполнения брешей, удаления поврежденной ДНК или вторичных структур, окружающих разрыв. Данный этап в процессе НГСК считают ответственным за случайную потерю нуклеотидов, связанную с репарацией НГСК. Один ключевой фермент процессинга концов при НГСК млекопитающих представляет собой Artemis, член суперсемейства ферментов металло-β-лактамаз, ген которого оказался мутированным у большинства пациентов с радиочувствительным тяжелым комбинированным иммунодефицитом (SCID) (Moshous и др., 2001). Artemis обладает как 5'→3' экзонуклеазной активностью, так и ДНК-ПКкс-зависимой эндонуклеазной активностью по отношению к ДНК, содержащей дн-он переходы (переходы от двух нитей к одной), и шпилькам ДНК (Ма и др., 2002). Его активность также регулируется протеинкиназой ATM. Таким образом, похоже, что Artemis, вероятно, участвует во множестве реакций на повреждение ДНК. Тем не менее, похоже, что Artemis участвует в репарации лишь подгруппы повреждений ДНК, так как не наблюдали значительного дефекта репарации ДНР в клетках, лишенных Artemis (Wang и др., 2005, Darroudi и др., 2007).
Бреши в ДНК должны быть заполнены, чтобы репарация стала возможной. Добавление нуклеотидов в ДНР могут осуществлять только полимеразы μ и λ (Lee и др., 2004; Сарр и др., 2007). Посредством взаимодействия с XRCC4 полинуклеотидкиназа (PNK) также привлекается к концам ДНК, чтобы позволить полимеризацию, а также лигирование ДНК (Koch и др., 2004). Наконец, НГСК завершается путем лигирования концов ДНК, этапа, осуществляемого комплексом, содержащим XRCC4, ДНК-лигазу IV и XLF (Grawunder и др., 1997). Другие лигазы могут частично заменять ДНК-лигазу IV, так как НГСК может происходить в отсутствие XRCC4 и лигазы IV (Yan и др., 2008). Более того, исследования показали, что XRCC4 и лигаза IV не играют роли вне НГСК, тогда как, напротив, KU принимает участие в других процессах, таких как транскрипция, апоптоз и реакции на микроокружение (Monferran и др., 2004;  и др., 2005; Downs и Jackson, 2004).
и др., 2005; Downs и Jackson, 2004).
НГСК можно ослабить или прекратить различными способами, многие из которых непосредственно затрагивают упомянутые выше белки (например, гетеродимер Ku80/Ku70, ДНК-ПКкс, но, в особенности, ДНК-лигазу IV, XRCC4, Artemis и XLF (XCC4-подобный фактор; или Cernunnos), PIKK (фосфоинозитид-3-киназа-подобное семейство протеинкиназ).
ГР: основные механизмы
Гомологичная рекомбинация (ГР) представляет собой очень точный механизм репарации. Гомологичная хроматида служит в качестве матрицы для репарации разорванной нити. ГР происходит в фазах S и G2 клеточного цикла, когда сестринские хроматиды становятся доступными. Классическая ГР главным образом характеризуется тремя этапами: 1) удаление 5'-нуклеотидов с разорванных концов, 2) внедрение нити и обмен с гомологичным дуплексом ДНК и 3) расхождение промежуточных продуктов (интермедиатов) рекомбинации. Репарация ДНР может завершаться различными путями, в зависимости от способности осуществлять внедрение нити, которые включают путь зависимого от синтеза ДНК отжига нитей (ЗСОН), классическую репарацию двухнитевого разрыва (РДНР) (Szostak и др., 1983), вызванную разрывом репликацию (ВРР) и, в качестве альтернативы, путь однонитевого отжига (ОНО). Все механизмы ГР взаимосвязаны и включают много общих ферментативных этапов.
Первый этап из всех реакций ГР соответствует удалению 5'-конца разорванной нити ДНК нуклеазами с помощью комплекса MRN (MRE11, RAD50, NBN (ранее NBS1, синдром разрывов Неймеген 1)) и CtIP (CtBP-взаимодействующий белок) (Sun и др., 1991; White и Haber, 1990). Полученная в результате этого совокупность последовательностей с 3'-одноцепочечными ДНР может искать гомологичную последовательность. Внедрение гомологичного дуплекса осуществляют с помощью нуклеофиламента, состоящего из 3'-онДНК, покрытой белком рекомбиназой RAD51 (Benson и др., 1994). Для сборки RAD51-филамента требуется привлечение репликативного белка A (RPA), гетеротримерного онДНК-связывающего белка, вовлеченного в метаболические процессы ДНК, связанные с онДНК, у эукариотов (Wold, 1997, Song и Sung, 2000). Затем RAD51 взаимодействует с RAD52, который обладает кольцеобразной структурой (Shen и др., 1996), чтобы вытеснить молекулы RPA и способствовать загрузке RAD51 (Song и sung, 2000). Rad52 важен для процессов рекомбинации у дрожжей (Symington, 2002). Тем не менее, у позвоночных, похоже, скорее BRCA2 (белок чувствительности к раку молочной железы 2 типа), чем RAD52 играет важную роль во внедрении и обмене нитей (Davies и Pellegrini, 2007; Esashi и др., 2007). Взаимодействие RAD51/RAD52 стабилизируется путем связывания с RAD54. RAD54 также играет роль в созревании интермедиатов рекомбинации после образования D-петли (Bugreev и др., 2007). С другой стороны, BRCA1 (рак молочной железы 1) взаимодействует с BARD1 (ассоциированный с BRCA1 RING-домен 1) и ВАСН1 (гомолог 1 ВТВ и CNC), чтобы осуществить лигазную и геликазную активность репарации ДНР, соответственно (Greenberg и др., 2006). BRCA1 также взаимодействует с CtIP CDK-зависимым образом и подвергается убиквитинированию в ответ на повреждение ДНК (Limbo и др., 2007). Следовательно, BRCA1, CtIP и комплекс MRN играют роль в активации ГР-опосредованной репарации ДНК в S и G2 фазах клеточного цикла.
Внедрение нуклеофиламента приводит к образованию гетеродуплекса, называемого петлей замещения (D-петлей), и включает замещение одной нити дуплекса внедряющейся нитью и ее спаривание с другой нитью дуплекса. Затем несколько путей ГР могут завершить репарацию, используя гомологичную последовательность в качестве матрицы для замещения последовательности, окружающей ДНР. В зависимости от используемого механизма взаимные обмены (кроссинговеры) между гомологичной матрицей и разорванной молекулой ДНК могут быть или не быть связаны с репарацией ГР. Кроссинговеры могут приводить к значительным генетическим последствиям, таким как геномные перестройки или утрата гетерозиготности.
В гомологичной рекомбинации также участвуют пять паралогов Rad51: Xrcc2, Xrcc3, Rad51B, Rad51C, Rad51D (Suwaki и др., 2011). Паралоги Rad51 образуют два типа комплексов: один, названный BCDX2, содержит Rad51B, Rad51C, Rad51D и Xrcc2; другой содержит Rad51C и Xrcc3 (СХ3) (Masson и др., 2001). Предположили, что первый комплекс участвует в образовании и/или стабилизации комплекса Rad51-ДНК (Masson и др., 2001). Похоже, что роль второго комплекса состоит в миграции ветви и расщеплении структуры Холлидея (Liu и др., 2007).
Ранее сообщалось, что увеличение ГР по сравнению с НГСК (см. публикацию патента США 20120231449, которая полностью включена в данную заявку посредством ссылки) можно применять, чтобы усилить и/или способствовать экспрессии трансгена.
Настоящее изобретение направлено на уменьшение или выключение ГР. ГР можно уменьшить или выключить различными способами, многие из которых непосредственно затрагивают упомянутые выше белки (например, белки комплекса MRN (MRE11, RAD50, NBN (ранее NBS1, синдром разрывов Неймеген 1)) и CtIP (CtBP-взаимодействующий белок), RAD51, белок репликации A (RPA), Rad52, BRCA2 (белок чувствительности к раку молочной железы 2 типа), RAD54, BRCA1 (рак молочной железы 1), взаимодействующий с BARD1 (ассоциированный с BRCA1 RING-домен 1), ВАСН1 (гомолог 1 ВТВ и CNC)). Настоящее изобретение сосредоточено на получении молекул РНК, таких как миРНК, для достижения данной цели.
Опосредованное микрогомологией соединение концов (ОМСК)
Когда другие пути рекомбинации не справляются или не активны, ДНР могут подвергаться репарации с помощью другого допускающего ошибки механизма репарации, называемого опосредованным микрогомологией соединением концов (ОМСК). Данный путь все еще требуется полностью охарактеризовать, и иногда его также называют альтернативным соединением концов (альт-СК), хотя не ясно, действительно ли в основе данных двух процессов лежит один и тот же механизм. Наиболее отличительное свойство данного пути, которое отличается от НГСК, состоит в использовании микрогомологий по 5-25 п.о. в процессе выравнивания разорванных нитей ДНК (McVey и Lee, 2008).
ОМСК может происходить на любой стадии клеточного цикла и не зависит от основных факторов НГСК и ГР, т.е. генов Ku70, лигазы IV и Rad52 (Boboila и др., 2010; Yu и McVey, 2010; Lee и Lee, 2007; Ma и др., 2003). Вместо того, чтобы использовать для инициации ОМСК собственный набор белков, наиболее важные применяемые для инициации белки являются компонентами комплекса MRN (MRX в дрожжах), содержащего Mre11, Rad50 и Nbs1 (Xrs2 в дрожжах), также участвующего в первых этапах ГР (Ма и др., 2003). Помимо комплекса MRN многие другие факторы были предложены в качестве участников в ОМСК, например, СТВР-взаимодействующий белок (CtIP; Yun и Hiom, 2009), поли(АДФ-рибоза)-полимераза 1 (PARP1), комплекс лигаза III/Xrcc1, лигаза I (Audebert и др., 2004), ДНК-полимераза θ (Yu и McVey, 2010) и комплекс ERCC1/XPF (Ма и др., 2003). Тем не менее, еще многие другие белки принимают участие в данном процессе.
Предположили, что в отсутствие других белков, связывающися с концом ДНК (таких как Ku или Rad51), ДНР узнаются PARP1, который затем инициирует их репарацию посредством ОМСК (McVey и Lee, 2008). Процесс репарации, аналогично ГР, начинается с удаления нуклеотидов с 5'- к 3'-концу, которое открывает короткие участки гомологии на каждой стороне от разрыва. Данный этап процессинга осуществляется комплексом MRN и регулируется CtIP (Mladenov и Iliakis, 2011). Комплементарные участки (присутствующие во фрагментах 3'-онДНК) спариваются и некомплементарные фрагменты (флэпы) удаляются (Yu и McVey, 2010), вероятно, комплексом ERCC1/XPF. Бреши (если такие есть) затем заполняются полимеразой (например, ДНК-полимеразой θ или δ (Yu и McVey, 2010; Lee и Lee, 2007)) и разрывы соединяются лигазой I или комплексом лигаза III/Xrcc1.
В отсутствие участков непосредственной микрогомологии на концах ДНК, что наиболее часто встречается, более удаленный фрагмент подвергнутой репарации молекулы можно скопировать, применяя точную ДНК-полимеразу (например, полимеразу θ). Данный дуплицированный участок затем участвует в выравнивании концов ДНК, что приводит к образованию вставки в месте соединения. Данный более сложный вариант опосредованной микрогомологией репарации назвали синтез-зависимой ОМСК (СЗ-ОМСК) (Yu и McVey, 2010).
Хотя ОМСК считали альтернативным путем рекомбинационной репарации, было показано, что он очень эффективен в процессе рекомбинации переключения классов IgH в В-лимфоцитах (Boboila и др., 2010), позволяя предложить, что он может играть большую роль, чем просто быть запасным механизмом. Также возможно, что некоторые ДНР, например, несовместимые выступы или тупые концы (которые плохо поддаются репарации посредством НГСК и/или ГР), могут более эффективно репарироваться посредством ОМСК (Zhang и Paull, 2005).
В ТАБЛИЦЕ D перечислены некоторые из ключевых генов в каждом из трех путей, которые, следовательно, также являются ключевыми мишенями для влияния на каждый из трех путей (см. также публикацию патента США 20120231449, которая полностью включена в данную заявку посредством ссылки). В указанную таблицу также включены белки, участвующие в репарации ДНК, такие как MDC1 и MHS2. MDC1 необходим для активации в ответ на повреждение ДНК контрольной точки в S-фазе и контрольной точки фазы G2/M клеточного цикла. Тем не менее, MDC1 также функционирует в опосредованной Rad51 гомологичной рекомбинации путем удерживания Rad51 в хроматине.
"Нокдаун" в контексте настоящего изобретения означает, что экспрессия целевого гена снижается, например, на 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 90% или более. Полное подавление означает, что больше нет детектируемой экспрессии целевого гена. В таблице D также показаны результаты, полученные для некоторых мишеней для нокдауна. Для специалиста в данной области должно быть очевидно, что существуют вариации в последовательностях нуклеиновых кислот указанных мишеней, следовательно, варианты генов, в частности, варианты, последовательности которых идентичны на 80%, 90% или 95% последовательностям мишеней, входят в объем настоящего изобретения.
ПРОЦЕССИНГ БЕЛКА И МЕТАБОЛИЧЕСКИЕ БЕЛКИ
Данная категория белков, которые можно применять для клеточной метаболической инженерии, не принадлежит ни к пути секреции белка, ни к пути рекомбинации, но иным образом влияет на процессы, свойственные экспрессирующей клетке.
Белки процессинга белка или метаболические белки часто представляют собой ферменты, такие как шапероны (см. выше определения шаперонов), протеин-изомеразы, добавляющие сахар ферменты (например, сиалил- или гликозилтрансферазы) или фосфатазы, или белки, контролирующие уровень энергии клетки или функции митохондрий.
В ТАБЛИЦЕ А приведен перечень белков, которые экспрессировали (экспр.) и/или экспрессию которых "подавляли" (нокдаун, НД), под подзаголовком "Процессинг белка и метаболические белки".
РАЗРАБОТКА ВЕКТОРА
Среди невирусных векторов транспозоны особенно привлекательны благодаря их способности встраивать единичные копии последовательностей ДНК с высокой частотой в многочисленные локусы внутри генома хозяина. Сообщалось, что, в отличие от вирусных векторов, некоторые транспозоны преимущественно не встраиваются близко к клеточным генам, и они, таким образом, с меньшей вероятностью приводят к повреждающим мутациям. Более того, транспозоны легко получить и легко с ними обращаться, включая, как правило, донорную плазмиду, несущую транспозон, которая содержит перемещаемую ДНК, фланкированную последовательностями инвертированных повторов, и экспрессирующую транспозазу вспомогательную плазмиду или мРНК. Разработали несколько систем транспозонов для перемещения ДНК в различных линиях клеток, не препятствуя эндогенным копиям транспозона. Например, транспозон PiggyBac (РВ), изначально выделенный из металловидки серой, эффективно переносит целевую ДНК в различные клетки млекопитающих.
Эпигенетические регуляторные элементы можно применять, чтобы защитить перемещаемую ДНК от нежелательных эпигенетических эффектов, когда ее помещают рядом с трансгеном в плазмидные векторы. Например, предложили элементы, называемые участками прикрепления к матриксу (MAR), чтобы повысить встраивание в геном и транскрипцию перемещаемой ДНК, при этом предотвращая замалчивание гетерохроматина, примером которых является эффективный MAR 1-68 человека. Они также могут действовать как инсуляторы и, тем самым, предотвращать активацию соседних клеточных генов. Элементы MAR, таким образом, применяют, чтобы опосредовать высокий уровень продолжительной экспрессии в контексте плазмидных или вирусных векторов.
В данной заявке показано, что при правильной разработке вектора благоприятные свойства эпигенетических регуляторов, в частности, элементов MAR, можно комбинировать с таковыми у векторов на основе транспозонов.
Транспозоны и векторы на основе транспозонов согласно настоящему изобретению можно применять в клеточной метаболической инженерии, например, для экспрессии секреторных белков из различных секреторных путей, описанных в данной заявке. Они также особенно полезны, когда требуется несколько раундов внедрения перемещаемой ДНК. Это подтвердили, когда тестировали множество белков секреторного пути клетки, когда трансфекция множеством векторов и/или множество следующих друг за другом циклов трансфекции могут исчерпать доступный антибиотик или помешать другим способам селекции. Возможность быстро экспрессировать терапевтические белки без необходимости селекции антибиотиком также представляет особый интерес, например, когда нужно экспрессировать множество кандидатных терапевтических белков с целью скрининга, поскольку можно получить значительные количества белков из не подвергнутых селекции популяций клеток через 2-3 недели после трансфекции. В частности, содержащие MAR векторы, несущие транспозоны, таким образом, представляют собой перспективное дополнение к доступному на сегодняшний день арсеналу векторов экспрессии.
Выбранные экспериментальные подходы позволили (в противоположность подходам, в которых полагаются на анализы на основе антибиотиков) провести различие между эффектами, основанными на (1) числе копий перемещаемой ДНК, и эффектами, основанными на (2) уровни экспрессии перемещаемой ДНК.
MAR 1-68 был особенно эффективен, когда он был расположен посередине между ITR транспозона PiggyBac, так как он не снижал эффективность транспозиции. MAR Х-29 также хорошо работал на краях транспозона, не снижая эффективность транспозиции или экспрессии.
Примечательно, что степень MAR-опосредованной активации перемещенных генов была меньше, по сравнению с таковой для самопроизвольного встраивания плазмиды. Более того, уровень экспрессии, нормированный, например, на копии трансгена, был выше при применении транспозонов, чем полученный в результате самопроизвольного встраивания плазмид в отсутствие транспозазы. Данный эффект наблюдали независимо от размера конструкций, присутствия MAR или силы промотора. Этого будут ожидать, так как транспозиция часто может происходить в геномных локусах, относительно позволяющих экспрессию, например, благодаря тому, что структуры хроматина могут быть более доступны как для транспозазы, так и для факторов транскрипции. В этом отношении, предшествующие исследования позволили предположить, что транспозоны преимущественно могут встраиваться внутрь интронов генов, в промоторы или в геномные локусы с более низкой способностью к замалчиванию, хотя данное утверждение остается темой для обсуждения. В качестве альтернативы, совместное встраивание множества копий плазмиды в один и тот же геномный локус, которое происходит в результате событий самопроизвольного встраивания, может приводить к образованию гетерохроматина и к замалчиванию повторяемых последовательностей, которому будет препятствовать MAR, тогда как встраивание единственной копии транспозона может в меньшей степени способствовать такому опосредованному хроматином замалчиванию. Кроме того, встраивание транспозонов в множество независимых геномных локусов повышает вероятность того, что по меньшей мере одна копия встроилась в благоприятное геномное окружение и экспрессируется, тогда как обнаружили, что встраивание плазмиды происходит преимущественно только в один геномный локус.
Наиболее высокие уровни экспрессии перемещаемой ДНК, например, трансген/TER, получили для содержащего MAR транспозона, когда он был связан с сильным промотором. Неожиданно было обнаружено, что высокие уровни экспрессии можно получить при перемещении незначительного числа копий ДНК, например, не более чем 20, 15, 10 или 5. Если, тем не менее, удается добиться высокой продуктивности, меньшее количество встроенных, например, перемещаемых копий ДНК является преимуществом, так как благодаря этому снижается вероятность возникновения точечных мутаций в одном или в подгруппе указанных трансгенов, возникающих в результате спонтанных событий мутагенеза. Кроме того, опосредованные транспозазой события встраивания менее мутагенны, чем механизмы репарации и рекомбинации ДНК, вовлеченные в самопроизвольное встраивание плазмиды, которые могут приводить к встраиванию неполных или перестроенных копий трансгена.
Высокая эффективность встраивания в геном с помощью транспозона piggyBac (РВ) также полезна, когда количество целевых клеток ограничено, например, для невирусного переноса терапевтических генов в первичные стволовые клетки для получения клональных популяций, например, для основанных на клеточной системе методов лечения или регенеративной медицины. В данном контексте, физиологические уровни экспрессии с нескольких перемещенных копий ДНК и частое возникновение событий транспозиции, таким образом, избавляющие от необходимости селекции антибиотиком, являются преимуществом, поскольку использование генов устойчивости к антибиотикам и/или ненадежной, например, экспрессии трансгена может вызывать сомнения относительно безопасности.
Влияние включения MAR на эффективность транспозиции
Так как устойчивость к антибиотику не обязательно отражает эффективную экспрессию трансгена, в качестве индикатора использовали зеленый флуоресцентный белок (GFP), экспрессируемый с производного сильного клеточного промотора GAPDH. Для того, чтобы проверить, может ли добавление элемента MAR в транспозон РВ влиять на эффективность транспозиции и экспрессию трансгена, и чтобы оценить, оказывает ли расположение MAR в конструкции какое-либо влияние на данные эффекты, разработали серию донорных конструкций, несущих транспозоны, содержащих GFP и ген устойчивости к пуромицину (Puro), в которые встроили MAR 1-68 или контрольную нейтральную спейсерную последовательность ДНК в различные положения в указанной плазмиде (фигура 1). Исходную несущую транспозон плазмиду Puro-GFP без вставки использовали в качестве контроля транспозиции, чтобы отличить вклад увеличенного размера транспозона от влияния добавления MAR или спейсерной последовательности.
В присутствии транспозазы наблюдали наиболее высокий уровень экспрессии GFP в не подвергнутых отбору клетках, когда MAR был расположен посередине, но не тогда, когда MAR помещали в 5'-3' направлении от последовательности, кодирующей GFP, и не тогда, когда его вставляли вне перемещенной последовательности, как и ожидалось (фигура 3А). При селекции пуромицином MAR-опосредованная активация уменьшалась, как с транспозазой, так и без нее, тогда как средние значения экспрессии GFP были повышены на один порядок величины (фигура 3В). Полученные рзультаты подтвердили, что отбор пуромицином позволял получить лишь небольшое число клеток, в которых уровни экспрессии наиболее высоки, что предполагали выше по подсчету событий транспозиции. Дополнительно показано, что векторы на основе транспозонов, содержащие MAR, расположенный по центру, позволяли получить уровни экспрессии, аналогичные таковым для такой же плазмиды, трансфицированной без транспозаз.
Влияние включения MAR на число копий встроенного транспозона
Более высокие уровни флуоресценции GFP могут возникать в результате повышенной транскрипции трансгенов и/или встраивания большего числа копий трансгена. Это оценивали путем определения количества встроенных в геном копий трансгена, внедренных с помощью различных типов векторов. Общую геномную ДНК выделяли из объединенных популяций клеток, либо после цитофлуориметрической сортировки флуоресцирующих клеток из не подвергнутых селекции популяций, либо после отбора по устойчивости к пуромицину. Число копий трансгена определяли с помощью анализа методом количественной полимеразной цепной реакции (qPCR) последовательности, кодирующей GFP, по сравнению с клеточным геном β2-микроглобулина (В2М). В отсутствие отбора антибиотиком среднее количество встроенных трансгенов, либо транспозазой, либо клеточными ферментами рекомбинации, было сходным, около 1-6 копий на геном, и на него значительно не влияли MAR или контрольная последовательность (фиг. 4А). Тем не менее, наименьшее число копий получали, когда MAR вставляли на границе транспозона, что поддерживает наше более раннее заключение, что он уменьшает транспозицию в данном положении. После селекции пуромицином клеток с высоким уровнем экспрессии количество перемещенных трансгенов оказалось в аналогичном диапазоне 2-7 копий (фиг. 4В). Тем не менее, количество копий трансгенов, встроенных в отсутствие транспозазы, как правило, было значительно выше и находилось в диапазоне от 6 до 14 копий. Это можно легко объяснить тем фактом, что самопроизвольное встраивание обычно приводит к встраиванию конкатемеров нескольких копий плазмиды в один геномный локус (результаты не представлены), и что большее число копий трансгенов должно приводить к более высоким уровням экспрессии, если клетки, подвергнутые влиянию замалчивания, были удалены путем отбора антибиотиком. Наряду с предшествующим заключением, что отбор антибиотиком позволяет получить преимущественно клетки с высоким уровнем экспрессии, это также свидетельствует о том, что самопроизвольное встраивание плазмиды приводит к большей вариабельности числа копий трансгена, чем при применении векторов на основе транспозонов.
Уровень экспрессии GFP затем нормировали на число копий гена, чтобы оценить внутренний экспрессионный потенциал указанных векторов независимо от их способности встраиваться в геном. В целом, получали более низкий уровень экспрессии на копию трансгена для не подвергнутых селекции клеток или для подверпнутых селекции антибиотиком клеток, трансфицированных без транспозазы или с MAR в центральном положении, что указывает на то, что на экспрессию трансгена влияет как включение эпигенетического регуляторного элемента, так и способ встраивания трансгена (фиг. 5). Уровень экспрессии на копию гена, как правило, повышался при применении транспозазы, когда проводили оценку различных векторов и комбинаций элементов, и данный эффект наблюдали как при отборе антибиотиком, так и без него. Наиболее высокие уровни экспрессии на копию трансгена получали после отбора антибиотиком клеток, в которые был внедрен вектор, несущий транспозон, содержащий элемент MAR в центральном положении, в присутствии транспозазы. Включение MAR непосредственно в 5'-3' направлении от последовательности, кодирующей GFP, значительно не повышало экспрессию трансгена, что было отмечено ранее для абсолютных уровней экспрессии.
Наконец, оценивали, может ли благоприятное влияние MAR 1-68 на экспрессию быть ограничено использованием сильного промотора GAPDH человека, применяемого в данном случае, или оно также будет иметь место с другими промоторами. Таким образом, мы заменили промотор GAPDH человека, запускающий экспрессию GFP, на более слабый ранний промотор вируса обезьян 40 (SV40). Применение более слабого промотора позволяло получить сравнимое количество GFP-положительных клеток и встроенных трансгенов, указывая на то, что экспрессия трансгена не изменяет эффективность транспозиции (не представленные результаты, фиг. 2А и фиг. 4В). Тем не менее, абсолютные уровни экспрессии были ниже при применении промотора SV40 (не представленные результаты по сранению с фиг. 3В). Кроме того, уровень экспрессии, нормированный на число копий транспозона, снижался в 4,6 раз при применении промотора SV40 в отсутствие MAR и в 3,1 раза в присутствии MAR 1-68 (результаты не представлены). Это свидетельствует о том, что MAR мог частично, но не полностью, предотвращать снижение экспрессии, возникшее в результате применения более слабого промотора, даже в присутствии транспозазы. В целом, можно показать, что нескольких встроенных копий достаточно для получения высокого уровня экспрессии трансгена с помощью транспозонов, и что наиболее высокий уровень экспрессии на трансген получали, когда, в данном контексте, MAR-68 помещали в 3'-5' направлении от сильного промотора.
Клетки СНО-М подвергали электропорации один или два раза одним вектором на основе транспозонов, содержащим трансген MAR Х_29. Эффективность транспозиции была наиболее высокой после электропорации (стабильная экспрессия наблюдалась в 30%-45% клеток). Тем не менее, уровни экспрессии трансгена были аналогичны таковым при химической трансфекции, при которой наблюдали меньшее количество положительных клеток и, следовательно, более низкую эффективность транспозиции. На фиг. 7 показаны результаты для легких и тяжелых цепей терапевтических иммуноглобулинов, встроенных в 3'-5' направлении от MAR Х_29, и были получены титры в диапазоне от 1 до 8 мкг/мл. Указанные уровни дополнительно повышались до 23-55 мкг/мл после сортировки экспрессирующих клеток (фиг. 7С).
Экспрессию трансгенов также можно существенно повысить, часто независимо от применения транспозонов, путем разработки определенного вектора, в частности, с применением определенного элемента(ов) MAR в определенных положениях относительно трансгена и, предпочтительно, комбинации данного элемента(ов) MAR с промоторами, энхансерами или продуктами их слияния.
Соответствующий вектор может содержать элементы MAR, которые фланкируют кассету экспрессии трансгена. Например, указанный вектор может содержать, например, элементы MAR (один или более), расположенные в 3'-5' направлении, и элементы MAR (один или более), расположенные в 5'-3' направлении, например, один MAR, расположенный в 3'-5' направлении от кассеты экспрессии трансгена, и один MAR, расположенный в 5'-3' направлении от нее (фиг. 18А, фиг. 18В, фиг. 19). Указанный вектор может содержать встроенный ген устойчивости к пуромицину под контролем промотора SV40. Трансген может находиться под контролем промотора GAPDH человека, слитого с энхансером немедленно-ранних генов цитомегаловируса (CMV) человека (в частности, слитый промотор CGAPD, обсуждаемый выше).
Наиболее высокий процентиль клеток с высоким и очень высоким уровнем продукции (% М3/М2), который оценивали по флуоресценции GFP с помощью анализа FACS и наименьшей вариабельности, можно было получить при применении 1_6R2, 1_68R2 и X_29R3 в качестве MAR, расположенных в 3'-5' направлении (свыше 80%, 80% и свыше 80%). Таким образом, процентили клеток с высоким и очень высоким уровнем продукции (% М3/М2), составляющие более чем 70%, более чем 75% или более чем 80%, входят в объем настоящего изобретения. Для специалиста в данной области должно быть очевидно, что допустимы некоторые отклонения от определенной последовательности данных элементов MAR. Соответственно, векторы, содержащие последовательности нуклеиновых кислот, более чем на 80%, 85%, 90%, 95% идентичные последовательностям SEQ ID NO: 6, 7, 8, 9 и 10, входят в объем настоящего изобретения (фиг. 18А, фиг. 18В).
Затухание экспрессии в биореакторе и/или в отсутствие селекционного давления часто ограничивает получение интересующего белка. Векторы, содержащие производные 1_68R2, 1_6R2 и X_29R3 MAR в качестве MAR, расположенных в 3'-5' направлении, исследовали на протяжении периода культивирования 5 недель в отсутствие селекции, и флуоресценцию GFP оценивали еженедельно в течение данного периода. При рассмотрении процентиля субпопуляции М3 обнаружили, что элемент 1_6R2 в качестве MAR, расположенного в 3'-5' направлении, и неперестроенный MAR 1-68 в качестве MAR, расположенного в 5'-3' направлении, оказались наилучшей из исследованных комбинаций в векторах с по меньшей мере одним элементом MAR, расположенным в 3'-5' направлении, и одним элементом MAR, расположенным в 5'-3' направлении (значительно выше 80% после более 2, 3, 4 недель) (см. фиг. 19).
Разработанный аналогичным образом вектор также может содержать, например, только элементы MAR (один или более), расположенные в 5'-3' направлении, например, один MAR, расположенный в 5'-3' направлении от кассеты экспрессии трансгенов (фиг. 20А, 20В, 21 и 22), и не содержать MAR, расположенного в 3'-5' направлении. Указанный вектор также, в данном случае, может содержать встроенный ген устойчивости к пуромицину под контролем промотора SV40. Трансген может находиться под контролем промотора GAPDH человека, слитого с энхансером немедленно-ранних генов цитомегаловируса (CMV) человека. См., например, SEQ ID NO: 11 и другие последовательности, идентичные более чем на 80%, 85%, 90% или 95% указанной последовательности. В такой комбинации с единичным MAR, где в качестве MAR выступал Х_29, добились превосходных результатов. Процентиль клеток с высоким уровнем экспрессии GFP (определенная, как описано выше), а также стабильность экспрессии с течением времени (определенная, как описано выше) были лучше, чем, например, таковые для векторов с высоким уровнем экспрессии, в которых элементы MAR фланкировали кассету экспрессии трансгена, а именно, для вектора, содержащего MAR 1_6R2, расположенный в 3'-5' направлении, и неперестроенный MAR 1-68, расположенный в 5'-3' направлении (см. фиг. 22). Данное открытие противопоставляется общепринятым предположениям, что элементы MAR наиболее эффективны, когда они фланкируют трансген (см. патент США 5731178). Стабильность экспрессии означает, что интересующая ДНК, например, трансген, экспрессируется популяцией клеток даже после некоторого периода времени, например, после более чем 4, 5, 6, 7, 8, 9, 10,11, 12,13 или 14 недель, на уровне, сравнимом (не более чем на 20%, 10 или 5% меньшем) и, особенно, даже более высоком, чем в течение двух недель от начала экспрессии. Часто стабильная экспрессия связана с высокой процентилью (например, более чем 80%) субпопуляций клеток с высоким уровнем экспрессии.
КЛЕТОЧНАЯ МЕТАБОЛИЧЕСКАЯ ИНЖЕНЕРИЯ: СЕКРЕТОРНЫЕ БЕЛКИ
Секреция гетерологичных белков, таких как IgG, снижается при неправильном процессинге полипептида и низкой продукции IgG в культивируемых клетках, таких как клетки СНО.
Наблюдали, что вызывалась экспрессия индуцированных стрессом шаперонов, таких как BiP, и что шаперон правильно локализовался в ЭР и был способен взаимодействовать с цепями предшественника IgG. Впрочем, IgG, содержащие определенные вариабельные последовательности, такие, которые обнаруживаются в инфликсимабе, тем не менее, неправильно процессировались и собирались, что приводило к низкой секреции. Следовательно, активация реакции несвернутых белков (UPR) в данных клетках оставалась неэффективной для восстановления существенного уровня иммуноглобулина.
Было показано, что SRP14 вовлечен в молекулярный этап секреторного пути, который является лимитирующим в клетках СНО, экспрессирующих сверх меры экзогенный белок. Примечательно, что данный лимитирующий этап также наблюдается для легко экспрессируемого трастузумаба, что легко приводит к получению клонов с высоким уровнем экспрессии. Данное заключение следовало за открытием, что экспрессия SRP14 человека легко восстанавливала экспрессию LP клонов, но она также повышала секрецию легко экспрессируемого IgG. Обнаружили, что экспрессия SRP14 повышает процессинг и доступность предшественников LC и НС и позволяет получить сравнимые уровни секреции обоих типов IgG. В целом, продемонстрировали, что SRP14 может быть, как правило, лимитирующим, когда секретируемые белки, такие как IgG, чрезмерно экспрессируются в клетках СНО.
Сильный эффект, полученный при экспрессии SRP14 в клетках СНО, наблюдаемый в данном исследовании, был неожиданным и позволил предположить, что SRP14 вызывает длительную задержку элонгации LC в клонах, с трудом продуцирующих IgG (фиг. 12В, пункт 1).
Предшествующее исследование показало, что сигнальный пептид, который выходит из транслирующей рибосомы, первый взаимодействует с субъединицей SRP54 частицы SRP, тогда как ассоциация с SRP9 и SRP14 может блокировать дальнейшую элонгацию образующегося полипептида, что приводит к остановке трансляции (Walter и Blobel 1981). На втором этапе комплекс рибосома-образующийся полипептид-SRP стыкуется с мембраной ЭР посредством взаимодействия с рецептором SR (Gilmore и др., 1982; Walter и др., 1982). SR затем может координировать высвобождение SRP из комплекса рибосома-образующийся полипептид и ассоциацию сайта выхода из рибосомы с каналом транслокона, через который растущий образующийся полипептид проникает в ЭР (Lakkaraju и др., 2008). Затем сопряженное с трансляцией перемещение может возобновиться, что приводит к удалению сигнального пептида и к синтезу правильно процессированных и секретируемых полипептидов.
Для правильного процессинга с трудом поддающихся экспрессии IgG может потребоваться необычно длинная трансляционная пауза, если кинетика стыковки с ЭР может быть медленнее для конкретных комбинаций последовательностей вариабельного домена IgG и сигнального пептида, вследствие неблагоприятных структур образующегося пептида. Таким образом, считали, что, в свою очередь, модуляция кинетики остановки трансляции путем экспрессии экзогенного компонента SRP14 человека улучшает правильную стыковку с ЭР и перемещение пре-LC в ЭР и, таким образом, восстанавливает эффективный процессинг сигнального пептида (фиг. 12В, пункт 2). Соответственно, снижение уровней SRP14 в клетках человека приводит к отсутствию задержки элонгации трансляции в полисомах, что может привести к чрезмерному увеличению длины образующихся полипептидов сверх критического значения, после которого SRP больше не сможет правильно нацелить секретируемый белок на ЭР.
Таким образом, аффинность SRP14 и возможно также SRP54 в клетках СНО к сигнальным последовательностям гетерологичного белка инфликсимаба человека будет снижена, что приведет к неправильной стыковке с ЭР и/или к элонгации образующегося пептида до того, как произойдет правильная стыковка. Это будет исправлено путем сверхэкспрессии SRP14 человека, увеличения периода времени, в течение которого остановленный комплекс рибосома-SRP может искать правильно организованный сайт стыковки на ЭР, несмотря на 'молекулярный затор' из сверхэкспрессированных белков IgG, происходящий на входе в ЭР.
Соответственно, сверхэкспрессия SR и транслокона, которые могут повышать вместительность ЭР в отношении транслокации, также приводила к улучшению секреции, даже в отсутствие сверхэкспрессии SRP14 человека. Наконец, продемонстрировали, что метаболическая инженерия секреторного пути посредством коэкспрессии комбинаций SRP человека, транслокона и субъединиц SR, приводит к дополнительному улучшению способности клетки секретировать белок, позволяя получить даже более высокие уровни секреции. В целом, пришли к заключению, что белки SRP, их рецепторы и транслокон могут быть, как правило, лимитирующими, когда секретируемые белки, такие как иммуноглобулины человека, чрезмерно экспрессируются в клетках СНО.
Немногое известно об относительном содержании SRP и компонентов мембраны ЭР по сравнению с секретируемыми белками и рибосомами в различных типах клеток, но в клетках, экспрессирующих большие количества рекомбинантного белка, потенциально могут возникать нарушения транслокации. Например, SR и/или транслокон могут стать лимитирующими, когда секретируемые белки экспрессируется на аномально высоких уровнях, или SRP14 может встречаться в клетках СНО на субстехиометрических уровнях по сравнению с другими субъединицами SRP. Соответственно, SRP9 и SRP14 присутствуют в 20-кратном избытке по сравнению с другими белками SRP в клетках приматов, но не в клетках мышей, и сверхэкспрессия SRP14 человека в нормальных клетках человека не повышает эффективность секреции щелочной фосфатазы. Более того, SRP14 человека крупнее, чем SRP14 грызуна, так как он содержит аланин-богатый хвост на С-конце, который не обнаружен в SRP14 грызуна. Таким образом, включение более крупного SRP14 человека в SRP СНО может приводить к образованию функциональной химеры SRP с более высокой активностью, с доминирующе-положительным эффектом.
Открытие того, что экспрессия цитозольных компонентов SRP, таких как SRP14, приводит к эффективному процессингу и секреции сверхэкспрессированных белков в клетках СНО, указывает на затруднение, которое можно использовать для увеличения выхода рекомбинантного белка. Данное затруднение ограничивает экспрессию различных и неродственных IgG и, возможно, также множества других моноклональных антител и производных, которые составляют несомненно наиболее распространенный класс рекомбинантных терапевтических белков.
Анализ интермедиатов секреции и возможных стрессовых реакций клетки с последующим систематическим поиском вышележащих лимитирующих действующих факторов, которые вызывают такую стрессовую реакцию, а затем, в конечном счете, разработкой метаболизма секреции клеток СНО привел к лучшему пониманию метаболических ограничений в данных клетках и того, как их обходить.
Гетерологичная экспрессия SRP14 восстанавливает секрецию и процессинг LC
Клоны IgG HP (с высоким уровнем продукции) и LP (с низким уровнем продукции) котрансфицировали с вектором, кодирующим компонент SRP14 SRP, и с плазмидой устойчивости к неомицину. Отдельные клетки из устойчивой к неомицину популяции клеток отделяли с помощью метода серийных разведений, а затем исследовали их рост и секрецию иммуноглобулина в партии чашек для культивирования при взбалтывании. Экспрессирующие SRP14 полученные из клонов LP субклоны секретировали значительно более высокие количества антитела, чем исходные клоны во всей культуре, и позволяли получить титры иммуноглобулина, аналогичные таковым для субклонов HP, экспрессирующих SRP14 (фиг. 8А и 8В, левые панели).
Экспрессия SRP14 не влияла на жизнеспособность клеток, но, похоже, она замедляла и продлевала рост культуры HP-клеток до аналогичных плотностей клеток (фиг. 8А и 8В, правые панели). Собрали культуральные супернатанты различных субклонов и анализировали в них концентрацию антитела. На фиг. 8С показано, что экспрессия SRP14 повышала секрецию из LP-клеток, что приводило к 7-кратному увеличению удельной продуктивности IgG. Более того, экзогенная экспрессия SRP14 также улучшала секрецию IgG из НР-субклонов, что приводило к 30% увеличению удельной продуктивности. Примечательно, что отдельные субклоны, экспрессирующие SRP14, секретировали трудно- и легкоэкспрессируемые IgG на по существу идентичных средних уровнях, при этом срединные значения удельных продуктивностей превышали 30 пикограмм на клетку в день (pcd). Такие очень высокие уровни секреции IgG поддерживались в течение более чем 6 месяцев культивирования, указывая на то, что это является постоянным свойством клеток, экспрессирующих SRP14.
Для того, чтобы дополнительно исследовать взаимосвязь между экспрессией SRP14 и продуктивностью в отношении IgG, анализировали уровни мРНК SRP14 в 5 отдельных экспрессирующих SRP14 LP-субклонах с помощью сравнительной количественной ПЦР. На фиг. 8D показано, что субклоны сверхэкспрессировали SRP14 на уровнях, находящихся в диапазоне от 50 до почти 200-кратных по сравнению с таковыми для эндогенных мРНК SRP14 в клетках СНО. Это сопровождалось улучшением секреции IgG в 4-6 раз по сравнению с LP контрольным клоном клетки. Примечательно, что наибольшую удельную продуктивность получили у субклона с промежуточным уровнем повышения экспрессии SRP14, приблизительно в 100 раз относительно уровня эндогенной мРНК SRP14 в клетках СНО (SRP14-LP субклон Е, результаты не представлены). Это подразумевает взаимозависимость уровня сверхэкспрессии SRP14 и удельной продуктивности IgG вплоть до порогового уровня SRP14, соответствующего 100-кратному повышению относительно эндогенного уровня экспрессии. Это позволило предположить, что другие компоненты секреторного пути, в свою очередь, могут стать лимитирующими при очень высоких уровнях SRP14, и что для оптимальной экспрессии IgG может быть необходима сбалансированная экспрессия компонентов данного пути.
Для того, чтобы проверить, может ли повышенная удельная продуктивность, полученная в процессе оценки клональной линии клеток, применяться в производственном процессе, лучшие экспрессирующие HP и LP субклоны SRP14 исследовали в чашках для культивирования при взбалтывании в условиях периодической культуры с подпиткой (т.е. LP субклон Е и HP субклон В, см. фиг. 8). Экспрессирующий SRP14 LP субклон позволял получить большие количества жизнеспособных клеток и титры иммуноглобулина, сходные с экспрессирующим SRP14 HP субклоном, при этом максимальная концентрация клеток составляла 8×106 клеток/мл и максимальный титр был выше 2 г на литр по окончании производственного цикла (фиг. 9А и В).
Затем исследовали вклад сверхэкспрессии SRP14 в синтез иммуноглобулина для двух данных субклонов. Результаты показали, что экспрессия SRP14 человека в субклоне, полученном из LP, позволяла получить нормально процессированную и зрелую LC, способную к фолдингу и сборке IgG (фиг. 10А, LP, дорожка S по сравнению с дорожкой -). Миграция свободной НС не изменилась, указывая на то, что экспрессия SRP14 действовала, в частности, на неправильно процессированную LC с трудом поддающегося экспрессии белка. Удивительно, что экспрессия SRP14 полностью нарушала накопление агрегировавшей LC в нерастворимой в Triton X фракции (фиг. 9А, нижняя панель). Экспрессия контрольного белка GFP не улучшала растворимость белка, а также не восстанавливала правильный процессинг LC (фиг. 10А, дорожка G для LP клеток). Экспрессия SRP14 не влияла на паттерн миграции НС и LC, полученный для HP субклона, и наблюдали небольшое влияние на количество свободных цепей и полностью собранного IgG по сравнению с контролями (фиг. 10А, дорожка G для HP клеток).
Осуществляли анализы с вытеснением на основе циклогексимида, чтобы исследовать кинетику фолдинга и сборки IgG, а также судьбу агрегатов IgG в экспрессирующем SRP14 субклоне, продуцирующем инфликсимаб. В противоположность исходным LP клеткам, содержащим агрегировавшие LC, неспособные участвовать в сборке IgG, экспрессирующий SRP14 LP субклон более не накапливал нерастворимые в Тх-100 LC (фиг. 9В, нижняя панель). Тем не менее, свободная LC оставалась в небольших количествах по сравнению со свободной НС, что также было отмечено для HP клеток, указывая на то, что она быстро включалась в состав HC-LC димеров и зрелого IgG и что она, возможно, ограничивала сборку IgG (фиг. 9А и 9В). В совокупности данные результаты позволяют предположить, что SRP14 может играть важную роль в процессинге LC в LP клетках, и что дополнительная экспрессия белка SRP может улучшить секрецию с трудом поддающихся экспрессии и легко поддающихся экспрессии IgG до аналогичных уровней.
Инженерия перемещения в ЭР улучшает секрецию рекомбинантного IgG
В связи с тем, что сверхэкспрессия гетерологичного SRP14 повышала секрецию IgG до данного порога, сделали вывод, что другие компоненты секреторного пути, которые взаимодействуют непосредственно или опосредованно с SRP14, могут стать лимитирующими в SRP14-LP субклонах. Следовательно, мы исследовали, может ли сверхэкспрессия других компонентов секреторного пути также улучшать экспрессию IgG, либо отдельно, либо в комбинации с SRP14. Данные компоненты включали белки SRP9 и SRP54 человека, которые входят в состав комплекса SRP вместе с SRP14, субъединицы рецептора SRP (SR) и субъединицы транслокона Sec61α, β и γ.
В первой серии экспериментов наилучший LP клон, а именно LP клон Е на фиг. 1, трансфицировали векторами экспрессии, кодирующими белки SRP или белки транслокона, отдельно или в комбинации. Затем оценивали продукцию IgG в полученных поликлональных популяциях LP клеток при периодическом культивировании. Экспрессия компонентов SRP или белков транслокона повышала секрецию иммуноглобулина данными повторно трансфицированными поликлональными популяциями LP клеток (фиг. 11А и не представленные результаты). По сравнению с экспрессией SRP14 отдельно сверхэкспрессия комбинаций белков SRP или транслокона улучшала удельную продуктивность трансфицированных поликлональных популяций LP клеток на дополнительные 20%-40% (фиг. 11А, сравнение срединных значений). Полученные результаты явно свидетельствуют о том, что определенные комбинации более эффективно восстанавливали секрецию инфликсимаба, чем экспрессия SRP14 отдельно, такие как комбинации, состоящие из экспрессии трех полипептидов SRP и его рецептора (SR), или из совместной экспрессии SR и транслокона (фиг. 11А).
Также оценивали, можно ли дополнительно оптимизировать экспрессирующий SRP14 LP субклон Е путем экспрессии комбинаций SR и/или транслокона. По сравнению с 30 pcd для SRP14-LP субклона Е, поликлональные популяции клеток, подвергнутые отбору после трансфекции белками SR и транслоконом, позволяли получить удельные продуктивности выше 60 pcd с трудом поддающегося экспрессии иммуноглобулина (фиг. 10В). Пришли к заключению, что векторы, экспрессирующие белки SR и транслокон, также можно применять для получения клонов с повышенными удельными продуктивностями по сравнению с SRP14-LP клоном E, и что можно успешно применять подход, основанный на сериях последовательных циклов трансфекции и селекции.
КЛЕТОЧНАЯ МЕТАБОЛИЧЕСКАЯ ИНЖЕНЕРИЯ: подавление пути рекомбинации и экспрессии ТЕР и функциональной РНК ТЕР
Для того, чтобы повлиять на рекомбинацию, можно подвергнуть замалчиванию важные гены рекомбинации ДНК. Мишенями для нокдауна являются гены, известные из литературы как важные для определенных путей рекомбинационной репарации у млекопитающих (таблица D). Поскольку большинство из данных генов необходимы для выживаемости и развития клетки, постоянное их замалчивание может привести к снижению жизнеспособности клетки. Следовательно, предпочтительно временное замалчивание данных целевых генов с применением РНК-интерференции (РНКи).
В клетках, обработанных смесью миРНК против факторов, вовлеченных в первые этапы НГСК, т.е. Ku 70, Ku80 и ДНК-ПКкс (один дуплекс миРНК на белок), частота событий НГСК была значительно ниже, чем в необработанных клетках. Взамен, в данных клетках ГР была более эффективной, что приводило к уменьшению отношения НГСК к ГР (от 2,7:1 до 1,3:1). Напротив, в клетках, обработанных миРНК, направленной против незаменимого фактора ГР - Rad51, ГР-зависимое восстановление уровня GFP почти полностью прекращалось, что повышало отношение НГСК : ГР до 5:1. Похоже, что данные результаты свидетельствуют о том, что репортерный анализ ГР и НГСК обладает достаточной чувствительностью, чтобы его можно было применять в экстрахромосомной форме. Они также дополнительно подвергают сомнению надежность используемых ранее мутантных линий клеток СНО (а именно, клеток 51D1, изначально опубликованных как ГР-отрицательные клетки, но, по-видимому, способных осуществлять ГР согласно данному анализу, результаты не представлены).
Повышение экспрессии и встраивания GFP в присутствии MAR. В параллельных экспериментах клетки СНО, обработанные различными миРНК, направленными против ГР или против НГСК, трансфицировали плазмидами, содержащими GFP или MAR-GFP. После двух недель отбора антибиотиком анализировали экспрессию и встраивание GFP в данных клетках с помощью FACS и количественной ПЦР, соответственно. При всех исследованных условиях добавление MAR приводило к 4-5-кратному повышению экспрессии GFP по сравнению с клетками, трансфицированными плазмидой без MAR (фиг. 13, фиг. 14А), что согласовалось с предшествующими сообщениями (Grandjean и др., 2011). Это повышение сопровождалось приблизительно 3-кратным увеличением числа копий встроенного GFP в клетках, трансфицированных плазмидой MAR-GFP, по сравнению с клетками, которые получили вектор без MAR (фиг. 13, фиг. 14В). Также наблюдалось 2-кратное увеличение средней флуоресценции GFP на копию гена (фиг. 13, фиг. 14С), возможно, вследствие более благоприятной локализации сайта встраивания (например, на участке, богатом эухроматином). Следовательно, можно предположить, что элементы MAR обладают способностью направлять гены в геномные локусы, позволяющие экспрессию гена. Комбинация миРНК против ГР с MAR (миРНК: RAD51, Rad51C и Brca1) приводила к изменению среднего уровня экспрессии GFP больше, чем в 11 раз, тогда как одна миРНК против ГР (миРНК: Rad51) приводила к изменению меньше, чем в 9 раз. Другое комбинации, такие как комбинация миРНК против ОМСК с MAR, также приводили к улучшенной экспрессии по сравнению с одной миРНК (результаты не представлены). Соответственно, комбинация различных миРНК (2, 3, 4, 5, 6, 8, 9, 10) к мишеням в том же или отличных метаболических путях, в частности, в пути рекомбинации, входит в объем настоящего изобретения.
Отсутствие влияния подавления гена НГСК на экспрессию и встраивание трансгена. Обработка миРНК против белков НГСК, похоже, значительно не влияла на стабильную экспрессию трансгена. Также не наблюдалось существенного изменения числа копий GFP в геноме по сравнению с необработанными клетками (за исключением клеток, обработанных миРНК против 53 ВР1, но только в отсутствие MAR, что, возможно, указывает на эффект, не свойственный рекомбинации посредством пути НГСК).
Повышение экспрессии и встраивания трансгена в отсутствие факторов ГР. В противоположность подавлению факторов НГСК, присутствие миРНК против белков ГР часто приводило к существенному повышению уровней стабильной экспрессии GFP по сравнению с необработанными клетками (за исключением Brca2, для которого наблюдалось существенное снижение уровня, но снова лишь в отсутствие MAR) (фиг. 15А). Как и в случае с замалчиванием генов НГСК, присутствие MAR приводило к повышению уровней стабильной экспрессии и встраивания GFP, а также к повышению уровней экспрессии на копию гена. На этот раз, тем не менее, замалчивание факторов ГР увеличивало данный эффект в 5-7 раз (наиболее ярким было подавление Rad51, приводящее к уровням экспрессии GFP, большим в 7,4 раза) (фиг. 15А), что может свидетельствовать о том, что белки ГР противодействуют положительному влиянию элемента MAR на экспрессию трансгена. В отсутствие MAR повышение экспрессии GFP коррелировало с увеличенным числом копий GFP в геноме (результаты не представлены). Неожиданно, этого не наблюдали в присутствии MAR, что указывает на то, что подавление белка ГР не влияло на опосредованное MAR увеличение числа копий (за исключением миРНК Rad51D). Похоже, это позволяет предположить, что отсутствие функционального пути репарации ГР не увеличивает количество событий рекомбинации, вызванных MAR. Вместо этого, оно стимулирует встраивание MAR и трансгена в более благоприятный локус, позволяя ему более эффективно экспрессироваться. Эта точка зрения также поддерживается повышенной экспрессией отдельных копий GFP в присутствии MAR в клетках, обработанных миРНК против ГР (фиг. 14С). Другая возможность состоит в том, что количество копий плазмиды, встроенных в геном, уже находится на максимальном уровне в контрольных клетках (при количестве плазмидной ДНК, используемом здесь), и его невозможно дополнительно увеличить даже в условиях, более благоприятных для MAR.
В совокупности, данные результаты позволяют предположить, что процесс опосредованного MAR встраивания трансгена преимущественно опосредован путем, противоположным гомологичной рекомбинации, хотя, вероятно, не путем НГСК, поскольку подавление его компонентов не влияло на встраивание или экспрессию. Можно предположить, что данный альтернативный путь менее активен в присутствии функционального пути ГР, но становится более важным, если ГР не работает.
ЭКСПРЕССИЯ кшРНК ПРОТИВ ГР ДЛЯ ПОВЫШЕНИЯ ЭКСПРЕССИИ ТЕРАПЕВТИЧЕСКИХ БЕЛКОВ
Три миРНК, нацеленные на Rad51, превратили в последовательности кшРНК, которые могут образовывать шпилечные структуры, и последовательность, кодирующую кшРНК, встраивали в вектор, несущий транспозон piggyBac, под контролем слитых энхансера GAPDH и промотора CMV, за которыми следовал MAR Х-29, но без гена селекции антибиотиком. Адаптированные для суспензии клетки СНО-М трансфицировали три раза донорной плазмидой, несущей транспозон, и вектором экспрессии транспозазы, после чего произвольно собирали 30 отдельных клонов клеток, применяя устройство ClonePix. Исходные клетки, а также экспрессирующую кшРНК популяцию клеток и клоны, повторно трансфицировали плазмидой, экспрессирующей GFP (а именно, конструкцией Puro-GFP-MAR Х_29), а затем осуществляли селекцию пуромицином поликлональных популяций экспрессирующих GFP клеток. Сравнение профилей флуоресценции GFP свидетельствовало о том, что получили более высокое соотношение клеток со средним-высоким уровнем флуоресценции (популяция клеток М2) или очень высоким уровнем флуоресценции (популяция клеток М3) из популяции клеток, трансфицированных вектором, несущим кшРНК, по сравнению с исходными клетками СНО-М (фиг. 17А).
Несколько клонов, экспрессирующих кшРНК, опосредовали очень высокие уровни GFP, при этом более 80% устойчивых к антибиотику клеток находились в субпопуляции МЗ с высоким уровнем флуоресценции через 10 дней после трансфекции, например, клон 16 и клон 26 (фиг. 17А и В). Высокие уровни флуоресценции GFP поддерживались в данных двух клонах после 35 дней дополнительного культивирования без селекции (фиг. 17С). Напротив, клон 17 не экспрессировал очень высокие уровни GFP в день 10, и экспрессия GFP, похоже, была нестабильной (фиг. 17В и С). Промежуточные уровни экспрессии и промежуточную стабильность получили для клона 8 и клона 22. Данные клоны также трансфицировали экспрессионными плазмидами, кодирующими легкую и тяжелую цепь с трудом поддающегося экспрессии терапевтического антитела инфликсимаба. По-прежнему, клоны 16 и 26 продуцировали наиболее высокие уровни антитела, за ними следовали клоны 8 и 22, тогда как клон 17 экспрессировал количества иммуноглобулина, которые были близки к 0,5-1 pcd, полученным для исходных клеток СНО-М (фиг. 17D и не представленные результаты). В клонах клеток, демонстрирующих наиболее значительно сниженные уровни мРНК Rad51, обнаруживали высокие уровни экспрессии как GFP, так и иммуноглобулина инфликсимаба, указывая на то, что повышенная экспрессия трансгена происходила в результате пониженной экспрессии рекомбинационного белка (результаты не представлены).
Таким образом, можно добиться более высоких уровней и более стабильной продукции секретируемых терапевтических белков, таких как инфликсимаб, клетками, экспрессирующими кшРНК, нацеленную на Rad51, или клетками, временно трансфицированными миРНК, такими как миРНК, нацеленная на Rad51.
МАТЕРИАЛЫ И МЕТОДЫ
Плазмиды и векторы ДНК
Вектор экспрессии, несущий транспозазу РВ, pCS2+U5V5PBU3, содержит последовательность, кодирующую транспозазу РВ, окруженную 5'- и 3'-нетранслируемыми концевыми участками (НТО) гена β-глобина Xenopus laevis. Данную плазмиду сконструировали, как описано далее: фрагмент 3'-НТО размером 317 п.о. из pBSSK/SB10 (любезно предоставленный доктором S. Ivies) встраивали в pCS2+U5 (Invitrogen/nife Technologies, Пейсли, Великобритания) с получением pCS2+U5U3. Последовательность, кодирующую транспозазу РВ (2067 п.о., номер доступа в GenBank: EF587698), синтезировали в ATG:biosynthetic (Мерцхаузен, Германия) и клонировали в каркас pCS2+U5U3 между двумя НТО. Контрольный вектор РВ соответствует немодифицированной плазмиде pCS2+U5 (фигура 1, левая панель).
Векторы, несущие различные транспозоны, используемые в данном исследовании, получали путем вставки 3'- и 5'-инвертированных концевых повторов (ITR) РВ размером 235 п.о. и 310 п.о., соответственно, синтезированных в ATG:biosynthetic (Мерцхаузен, Германия), в плазмиду pBluescript SK (pBSK ITR3'-ITR5', фигура 1, правая панель). Затем вставляли ген устойчивости к пуромицину (PuroR) под контролем промотора SV40 из плазмиды pRc/RSV (Invitrogen/Life Technologies) между двумя указанными ITR. Элементы MAR 1-68 и MAR Х-29, гены устойчивости к пуромицину и GFP, применяемые в данном исследовании, были такие, как описано ранее. Векторы экспрессии иммуноглобулинов и последовательности, кодирующие SRP9, SRP14, SRP54, SRPR-альфа, SRPR-бета, SEC61A1, SEC61B и SEC61G, были такие, как описано в Le Fourn и др. (Metab. Eng., Epub, 1 февраля 2013 г.)
GFP, иммуноглобулин или секреторные белки экспрессировали, применяя эукариотическую кассету экспрессии, состоящую из энхансера CMV человека и промотора GAPDH человека, расположенных в 3'-5' направлении от кодирующей последовательности, за которыми следовал сигнал полиаденилирования SV40, терминатор гастрина человека и энхансер SV40 (см. Le Fourn и др., 2013). Кассеты экспрессии и/или элементы MAR встраивали между последовательностями ITR или в каркас бактериального вектора, как показано на фигуре 1 и в описаниях к фигуре, применяя стандартные способы клонирования.
Культивирование клеток и анализ трансфекции
Линию клеток DG44 СНО культивировали в DMEM: F12 (Gibco), дополненной гипоксантином/тимидином (НТ, Gibco) и 10% фетальной бычьей сывороткой (FBS, Gibco). Трансфекцию осуществляли, применяя PEI (JetPRIME, Polyplus Transfection), следуя инструкциям производителя. Клетки трансфицировали различными количествами плазмидной ДНК, содержащей транспозазу РВ (в диапазоне от 0 до 1500 нг), для экспериментов с титрованием или котрансфицировали оптимальным соотношением 300 нг плазмиды, экспрессирующей транспозазу РВ, и 300 нг донорной плазмиды, несущей транспозон. Через два дня после трансфекции клетки переносили в несколько чашек Петри в зависимости от эксперимента. Для анализа не подвергнутых селекции трансфицированных клеток СНО, клетки пересевали без селекции антибиотиком в течение 3 недель и процент флуоресцентных клеток и интенсивность флуоресценции GFP-положительных клеток определяли с помощью анализа FACS, применяя проточный цитометр CyAn ADP (Beckman Coulter). Для анализа числа копий гена в не подвергнутых селекции клетках стабильные GFP-положительные клетки СНО подвергали сортировке, применяя FACSAriall. Для количественного анализа устойчивых к антибиотику колоний, 50000 трансфицированных клеток высевали в чашки диаметром 100 мм и подвергали селекции 5 мкг/мл пуромицина в течение 2 недель. Затем устойчивые колонии фиксировали и окрашивали 0,7% метиленовым синим в 70% EtOH в течение 10 мин, и подсчитывали колонии диаметром >0,5 мм. Для исследования экспрессии GFP клетки подвергали селекции в течение двух недель перед анализом FACS флуоресценции GFP, описанным выше.
Клетки СНО-М поддерживали в суспензионной культуре в бессывороточной среде SFM4CHO Hyclone (ThermoScientific), дополненной L-глутамином (РАА, Австрия) и добавкой НТ (Gibco, Invitrogen life sciences), при 37°C, 5% CO2 в увлажненном воздухе. Несущие транспозон донорные плазмиды переносили в данные клетки путем электропорации согласно рекомендациям производителя (Neon devices, Invitrogen). Проводили количественный анализ секреции иммуноглобулина периодическими культурами, как описано ранее (см. Le Fourn и др., 2013). Вкратце, популяции клеток, экспрессирующих иммуноглобулины, оценивали при периодическом культивировании в минибиореакционных пробирках объемом 50 мл (ТРР, Швейцария) при 37°С во влажной камере при 5% CO2 в течение 7 дней. Концентрации иммуноглобулина в культуральных супернатантах измеряли с помощью «сэндвич»-иммуноферментного анализа (ИФА).
Анализы числа копий генов методом количественной ПЦР (qPCR)
Общую ДНК выделяли из популяций стабильных клеток СНО после анализов транспозиции, применяя набор DNeasy Tissue (Qiagen, Хильден, Германия), следуя протоколу производителя. Число копий встроенных в геном трансгенов оценивали, применяя 6 нг геномной ДНК, с помощью количественной ПЦР, применяя набор с SYBR Green-Taq полимеразой от Eurogentec Inc и устройство для проведения ПЦР ABI Prism 7700 (Applied Biosystems). Праймеры GFP-прямой: ACATTATGCCGGACAAAGCC и GFP-обратный: TTGTTTGGTAATGATCAGCAAGTTG использовали для определения количества гена GFP, тогда как праймеры В2М-прямой: ACCACTCTGAAGGAGCCCA и В2М-обратный: GGAAGCTCTATCTGTGTCAA использовали для амплификации гена бета-2 микроглобулина. Для ампликона, полученного с помощью праймеров В2М, обнаружили одно совпадение на гаплоидный геном СНО после выравнивания с нашей сборкой генома СНО, применяя программное обеспечение NCBI BLAST. Так как СНО являются почти диплоидными клетками, приблизительно подсчитали, что В2М присутствует в 2 копиях на геном. Соотношения числа копий целевого гена GFP рассчитывали по сравнению с таковым для эталонного гена В2М, как описано ранее.
Сортировка и анализ экспрессирующих иммуноглобулин клеток
Для сортировки магнитным способом экспрессирующих IgG клеток, трансфицированные клетки СНО-М высевали при концентрации клеток 3×105 клеток в мл в среде SFM4CHO (Thermo Scientific), дополненной 8 мМ L-глутамином и добавкой 1×НТ (оба от Gibco), называемой полной средой. После 4 дней в культуре 2×106 клеток промывали, ресуспендировали в ФБР и инкубировали с биотинилированным IgG человека (KPL216-1006) в конечной концентрации 3 мкг/мл вместе с 30 мкл заранее промытых покрытых стрептавидином гранул MyOne Т1 Dynabeads (Invitrogen), на вращающемся колесе в течение 30 минут при комнатной температуре. Смесь клеток и гранул затем помещали на магнит, чтобы отделить помеченные клетки от не помеченных клеток. Гранулы промывали 4 раза фосфатно-солевым буферным раствором (ФБР). После последней промывки ФБР гранулы и клетки ресуспендировали в 500 мкл заранее нагретой полной среды, переносили в 24-луночный планшет и инкубировали при 37°С при 5% CO2. После 24 ч отделенные методом магнитной сортировки поликлональные клетки отделяли от гранул и продолжали инкубацию до тех пор, пока клетки не достигли достаточной плотности для их размножения в 50 мл центрифужных биореакторах ТРР (TECHNO PLASTIC PRODUCTS AG, Швейцария).
В качестве альтернативы, выделяли два клона из не подвергнутых сортировке и не подвергнутых селекции популяций, экспрессирующих каждый из трех IgG, применяя устройство clonePix. Вкратце, для иммобилизации отдельных клеток применяли полутвердые среды, и колонии, секретирующие большие количества IgG, собирали через десять дней после заливки. Данные линии клеток пересевали каждые 3-4 дня в центрифужные биореакторы при плотности 3×105 клеток/мл в содержащую пептон ростовую среду (SFM4CHO, дополненную 8 мМ глутамином, от Hyclone) и инкубировали во влажной камере при 37°С и 5% CO2 при вращении на 180 об/мин.
При оценке поликлональных популяций клеток титры IgG определяли для клеток, которые высевали при концентрации 1×105 клеток в мл и растили в течение 6 дней в 5 мл полной среды в 50 мл центрифужных биореакторах. В качестве альтернативы, культуры клональных популяций во встряхиваемых колбах инокулировали при плотности 3×105 клеток/мл в среде SFM4CHO, чтобы начать производственный процесс периодического культивирования с подпиткой. Производство периодических культур с подпиткой осуществляли в культуре объемом 25 мл во встряхиваемых колбах объемом 125 мл или в культуре объемом 5 мл в культуральных пробирках ТРР объемом 50 мл во влажных камерах при 37°С и 5% CO2 при взбалтывании при 150 об/мин (встряхиваемые колбы и центрифужные пробирки объемом 125 мл). Производство осуществляли в течение десяти дней с подпиткой 16% от исходного объема культуры концентрированной подпитки с определенным химическим составом (Hyclone, Cell Boost 5, 52 г/л) в дни ноль, три и шесть-восемь. В процессе культивирования не применяли подпитку глутамином и глюкозой. Жизнеспособность и концентрацию жизнеспособных клеток (VCD) в культуре измеряли ежедневно, применяя устройство GUAVA (Millipore). Двойной анализ «сэндвич»-ИФА применяли, чтобы определить концентрации МАТ, секретируемых в культуральные среды.
Плазмиды и относительный количественный анализ ПЦР
Клонирующие векторы, применяемые в данном исследовании, представляли собой векторы для экспрессии в млекопитающем SLXplasmid_082 от Selexis. Последовательность люциферазы контрольного вектора pGL3 (Promega) заменяли на эукариотическую кассету экспрессии, состоящую из энхансера CMV человека и промотора GAPDH человека, расположенную в 3'-5' направлении от последовательности, кодирующей EGFP, за которой следовал сигнал полиаденилирования SV40, терминатор гастрина человека и энхансер SV40. Два полученных из MAR генетических элемента человека фланкировали указанную кассету экспрессии, и ген устойчивости к пуромицину экспрессировался под контролем промотора SV40, при этом SLXplasmid_082 отличается по типу элемента MAR, расположенного в 3'-5' направлении от указанной кассеты экспрессии (hMAR 1-68 и hMAR X-29; Girod и др., 2007).
кДНК тяжелых и легких цепей трастузумаба и инфликсимаба клонировали в вектор экспрессии, чтобы заменить EGFP. Вектор, несущий кассеты экспрессии обеих тяжелой и легкой цепей каждого IgG, получали путем объединения кассет экспрессии тяжелой и легкой цепей на одном плазмидном векторе. Последовательности сигнальных пептидов из всех тяжелых и легких цепей, а также константные области легких цепей идентичны. Константные области тяжелых цепей отличаются в нескольких положениях аминокислот (варианты DEL по сравнению с ЕЕМ).
Праймеры для амплификации методом ПЦР и номера доступа в GenBank кДНК SRP9, SRP14, SRP54, SRPR-альфа, SRPR-бета, SEC61A1 и SEC61B перечислены в других местах в данном тексте. Продукты ПЦР, кодирующие секреторные белки, клонировали в вектор, чтобы заменить последовательность EGFP. Когда несколько секреторных белков экспрессировали совместно, инвертированные концевые последовательности транспозона piggyBac встраивали в векторы, чтобы заключить в них кассету экспрессии, и полученные в результате этого векторы котрансфицировали с вектором экспрессии, несущим транспозазу piggyBac, чтобы улучшить встраивание трансгена и избавиться от необходимости отбора антибиотиком.
Обычный вектор экспрессии, несущий транспозазу РВ, который представлял собой pCS2+U5V5PBU3, содержащий последовательность, кодирующую транспозазу РВ, окруженную 5'- и 3'-нетранслируемыми концевыми областями (НТО) гена β-глобина Xenopus laevis, применяли в смежных экспериментах. Данную плазмиду конструировали, как описано далее: фрагмент 3'-НТО размером 317 п.о. из pBSSK/SB10 встраивали в pCS2+U5 (Invitrogen/Life Technologies, Пейсли, Великобритания) с получением pCS2+U5U3. Последовательность, кодирующую транспозазу РВ (2067 п.о., номер доступа в GenBank: EF587698), синтезировали в ATG:biosynthetic (Мерцхаузен, Германия) и клонировали в каркас pCS2+U5U3 между двумя НТО. Контрольный вектор РВ соответствовал немодифицированной плазмиде pCS2+U5.
Векторы, несущие различные транспозоны, получали путем вставки 3'- и 5'-инвертированных концевых повторов РВ (ITR) размером 235 п.о. и 310 п.о., соответственно, синтезированных в ATG:biosynthetic (Мерцхаузен, Германия), в плазмиду pBluescript SK (pBSK ITR3'-ITR5'). Ген фосфотрансферазы неомицина (NeoR) под контролем промотора SV40 из плазмиды pRc/RSV (Invitrogen/Life Technologies) затем вставляли между двумя указанными ITR. Элементы MAR 1-68 и MAR Х-29, гены устойчивости к пуромицину и GFP, применяемые в данном исследовании, были такие, как описано ранее (Girod и др. 2007; Grandjean и др. 2011; Hart и Laemmli 1998). Векторы экспрессии иммуноглобулинов и последовательности, кодирующие SRP9, SRP14, SRP54, SRPR-альфа, SRPR-бета, SEC61A1 и SEC61B, описаны в данной заявке. Секреторные белки экспрессировали, применяя эукариотическую кассету экспрессии, состоящую из энхансера CMV человека и промотора GAPDH человека, расположенных в 3'-5' направлении от кодирующей последовательности, за которыми следовал сигнал полиаденилирования SV40, терминатор гастрина человека и энхансер SV40. Кассеты экспрессии и/или элементы MAR встраивали между последовательностями ITR или в каркас бактериального вектора, применяя стандартные способы клонирования.
Системы транспозона PiggyBac, включая подходящие 3'- и 5'-ITR, а также транспозазу, например, доступны для приобретения у SYSTEM BIOSCIENCE.
Для относительного анализа методом количественной ПЦР общую РНК выделяли из 1×105 клеток и транскрибировали с помощью обратной транскриптазы в кДНК, применяя набор FastLane Cell cDNA (Qiagen), следуя инструкциям производителя. Осуществляли количественный анализ экспрессии SRP14 и GAPDH с помощью количественной ПЦР, применяя Rotor Gene Q (Qiagen) и LightCycler®480 SYBR Green I Master (Roche), с применением праймеров, перечисленых в приложенной таблице S1. Уровни информационной РНК SRP14 нормировали на таковые для GAPDH, применяя программное обеспечение Rotor-Gene Q Series (Qiagen).
Культивирование клеток, стабильная трансфекция и субклонирование линий клеток СНО
Суспензионные клетки яичника китайского хомячка (CHO-K1) поддерживали в бессывороточной среде SFM4CHO Hyclone (ThermoScientific), дополненной L-глутамином (РАА, Австрия) и добавкой НТ (Gibco, Invitrogen life sciences), при 37°C, 5% CO2 в увлажненном воздухе. Клетки СНО-K1 трансфицировали векторами экспрессии тяжелых и легких цепей трастузумаба или инфликсимаба, несущими ген устойчивости к пуромицину, путем электропорации согласно рекомендациям производителя (Neon devices, Invitrogen). Через два дня клетки переносили в планшеты Т75 в среду, содержащую 10 мкг/мл пуромицина, и указанные клетки дополнительно культивировали в условиях селекции в течение двух недель. Затем получали стабильные отдельные клоны клеток, экспрессирующих IgG трастузумаб и инфликсимаб, с помощью метода серийных разведений, растили и анализировали эффективность их роста и уровни продукции IgG. Продуцирующие IgG трастузумаб и инфликсимаб клоны клеток, экспрессирующих наиболее высокие уровни IgG, отбирали для дополнительных биохимических экспериментов. Некоторые из данных клонов затем котрансфицировали путем электропорации вектором, экспрессирующим SRP14, и плазмидой, несущей ген устойчивости к неомицину. Клетки затем культивировали в среде, содержащей 300 мкг/мл G418, в течение двух недель, как описано выше. Стабильные клоны выделяли с помощью ограниченного разведения и подтверждали экспрессию SRP14 с помощью анализов методом количественной ПЦР перед выращиванием культуры для биохимического анализа.
Периодическое культивирование и периодическое культивирование с подпиткой
Эффективность роста и продукции для отдельных клонов, экспрессирующих трастузумаб и инфликсимаб, оценивали при периодическом культивировании в минибиореакторе объемом 50 мл (ТРР, Швейцария) при 37°С во влажной камере при 5% CO2 в течение 7 дней. В день 3, день 4 и день 7 культивирования клеток определяли концентрацию и жизнеспособность клеток, применяя систему для проточной цитометрии Guava EasyCyte (Millipore). Титр IgG в культуральных супернатантах измеряли с помощью «сэндвич»-ИФА. Плотность клеток (Cv*мл-1) и значения титра IgG (мкг*мл-1) наносили на график в указанный день отбора образцов в процессе эксперимента. Удельную продуктивность IgG клонов, экспрессирующих трастузумаб и инфликсимаб, определяли как падение концентрации IgG в зависимости от суммарного количества жизнеспособных клеток (IVCD), рассчитанное в день 3 - день 7 (фаза продукции), и выражали в виде пг на клетку в день (pcd).
Для периодических культур с подпиткой клетки высевали при плотности 0,3×106 клеток/мл во встряхиваемые колбы объемом 125 мл в 25 мл бессывороточной среды SFM4CHO Hyclone. Культуры поддерживали при 37°С и 5% CO2 при встряхивании. Культуры подпитывали ежедневно коммерчески доступной подпиткой Hyclone (ThermoScientific). Плотности клеток и продукцию IgG оценивали ежедневно.
Анализ экспрессии и агрегации белков
Растворимые цитоплазматические белки экстрагировали путем пермеабилизации клеток 1% Triton Х-100 в буфере ФБР в присутствии коктейля ингибиторов протеаз (Roche, inc). После инкубации в течение 30 мин на льду клетки центрифугировали 10 мин при 14000 об/мин. Супернатант назвали фракцией "растворимых цитозольных и ЭР белков". Осадок растворяли путем обработки ультразвуком в буфере Лэммли с мочевиной (62,5 мМ Трис, 2% ДСН, 8 М мочевина, 5% глицерин, краситель бромфеноловый синий), что позволяло получить нерастворимую фракцию агрегировавших и везикулярных белков. Растворимую и нерастворимую фракции затем разбавляли буфером Лэммли с добавлением или без 2-меркаптоэтанола и кипятили 8 мин при 95°С. Образцы в восстанавливающем и невосстанавливающем буферах разделяли на 10% или градиентном 4-10% акриламидных гелях с помощью электрофореза в полиакриламидном геле с добавлением додецилсульфата натрия (ЭФ в ПААГ/ДСН), соответственно.
Белки затем переносили на нитроцеллюлозную мембрану. После блокирования в 5% молоке, разбавленном в TBS-Tween (20 мМ Трис, 0,5 М NaCl, 0,1% Tween 20), мембраны анализировали для обнаружения различных белков, применяя следующие первичные антитела: антитело осла против IgG (H+L) человека, конъюгированное с пероксидазой хрена (JK immunoresearch, №709035149, 1:5000), поликлональные антитела кролика против BiP человека (Cell signalling, BiP, С50В12, 1:2000), моноклональное антитело мыши против CHOP человека (Cell signalling, CHOP, L63F7, 1:500), поликлональное антитело козы против GAPDH человека. После инкубации в течение ночи при 4°С каждую мембрану обрабатывали конъюгированными с пероксидазой хрена HRP IgG против антител кролика или IgG против антител мыши (Cell signalling, 1:20000). Белки, специфично узнаваемые каждым антителом, детектировали, применяя реагенты для улучшенной хемилюминесценции (ECL) и экспонирование с пленкой для ECL (Amersham Biosciences).
Эксперименты с вытеснением белков на основе циклогексимида
Эксперименты с вытеснением на основе циклогексимида осуществляли на клонах СНО-K1 с высоким (HP) и низким (LP) уровнем продукции IgG. Равные количества клеток высевали на 6-луночные планшеты в полной культуральной среде, дополненной 100 мкМ циклогексимида (Sigma). В различные моменты времени клетки собирали и лизировали в ФБР, 1% Triton Х-100. Растворимые и нерастворимые в Тх фракции затем разделяли на 4-10% акриламидном геле с помощью ПААГ/ДСН в невосстановительных условиях и подвергали иммуноблоттингу с антителом к IgG человека.
Анализы дифференциального фракционирования детергентом
Фракционирование цитозольных и мембранных белков осуществляли путем дифференциального экстрагирования детергентом из осадка клеток. Клетки сначала промывали в 1 мл ФБР, и плазматические мембраны HP и LP клеток пермеабилизовали в буфере КНМ (110 мМ KAc, 20 мМ HEPES, 2 мМ MgCl2, pH 7,2), содержащем 0,01% дигитонин (Sigma), в течение 10 мин в присутствие или без 1% Triton Х-100. Полупермеабилизованные клетки однократно промывали в буфере КНМ и добавляли трипсин до конечной концентрации 50 мкг/мл на 10 мин при комнатной, температуре, чтобы расщепить растворимые белки. Расщепление трипсином останавливали путем добавления 1 мМ фенилметилсульфонилфторида (PMSF) и 4 мМ 4-(2-аминоэтил)-бензосульфонилфторида (AEBSF). Клетки собирали путем центрифугирования и растворимые белки экстрагировали в присутствии Triton Х-100 и ингибитора протеаз, как описано а разделе 2.4. Восстанавливающий буфер Лэммли, содержащий 2-меркаптоэтанол, добавляли к осадку и фракциям супернатанта, которые затем подвергали ЭФ в 8% ПААГ/ДСН. Осуществляли иммуноблоттинг, чтобы детектировать белки IgG и BiP.
Перекрестное сшивание белков и анализ методом вестерн-блоттинга
Клетки, продуцирующие инфликсимаб на низком уровне (LP), однократно промывали в ФБР и инкубировали с добавлением или без 1 мМ кросслинкера дитиобис(сукцинимидилпропионата) (DSP) (ThermoScientific) в течение 30 мин на льду. Перекрестное сшивание гасили путем инкубации с добавлением 50 мМ Трис-HCl (pH 7,4) в течение 10 мин перед экстрагированием белка в буфере ФБР, содержащем 1% Triton Х-100. После центрифугирования в течение 10 мин при 14000 об/мин в микроцентрифуге нерастворимую в Triton Х-100 фракцию или цельный экстракт белка анализировали с помощью ЭФ в ПААГ/ДСН в восстановительных условиях, подвергали иммуноблоттингу и окрашивали антителами против BiP и LC. Равные количества белков из нерастворимых в Тх фракций анализировали параллельно.
Должно быть очевидно, что способы и композиции согласно настоящему изобретению могут быть включены в виде различных вариантов реализации, лишь незначительное количество которых описано в данной заявке. Для специалиста должно быть очевидно, что существуют другие варианты реализации, которые не выходят за рамки сущности настоящего изобретения. Таким образом, описанные варианты реализации являются иллюстративными и не должны истолковываться как ограничительные.
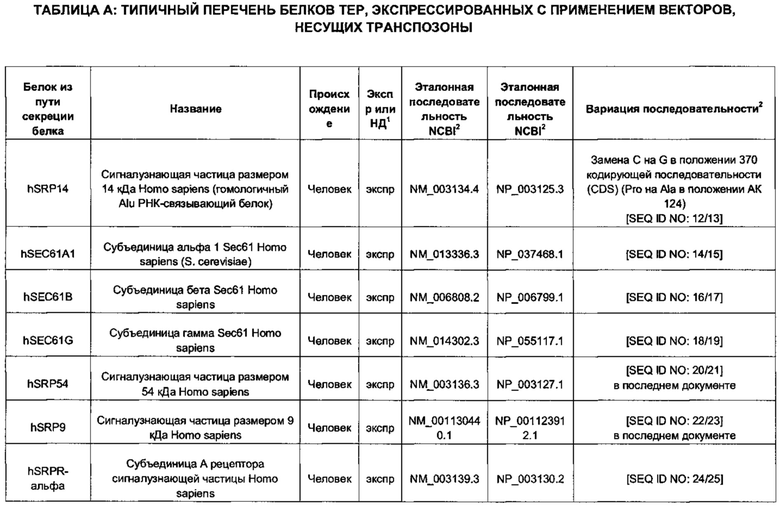
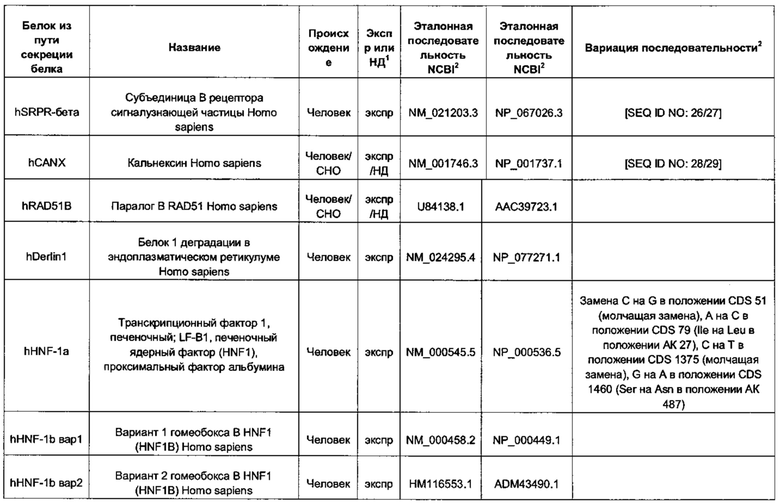
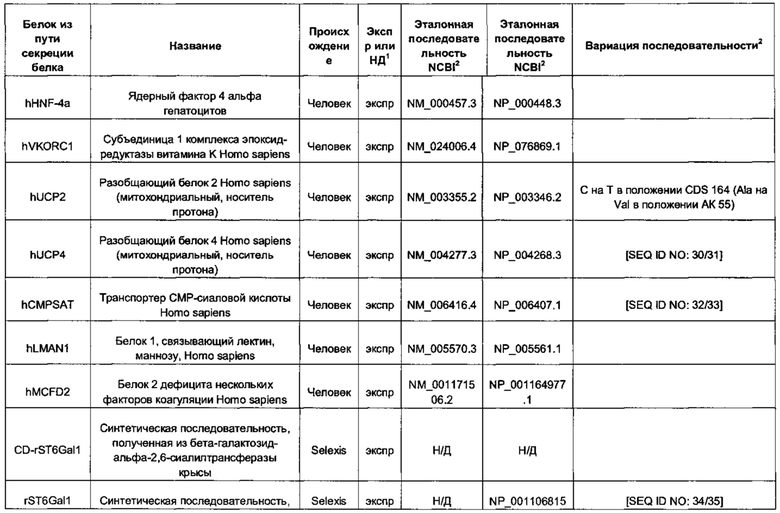
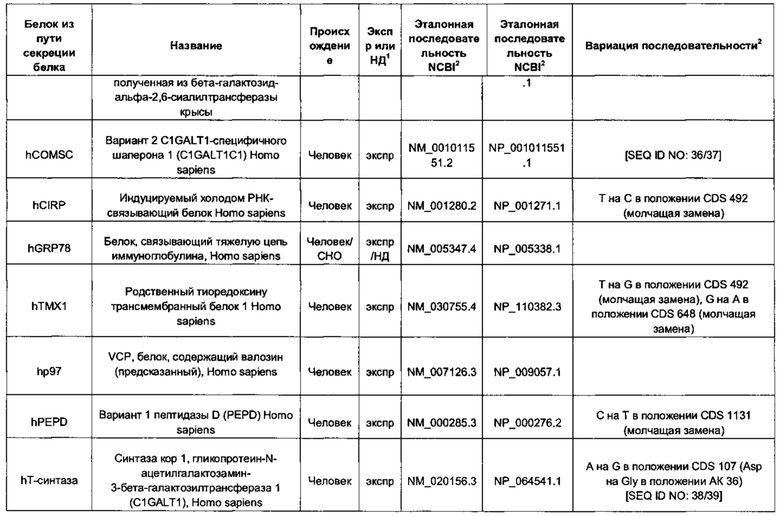
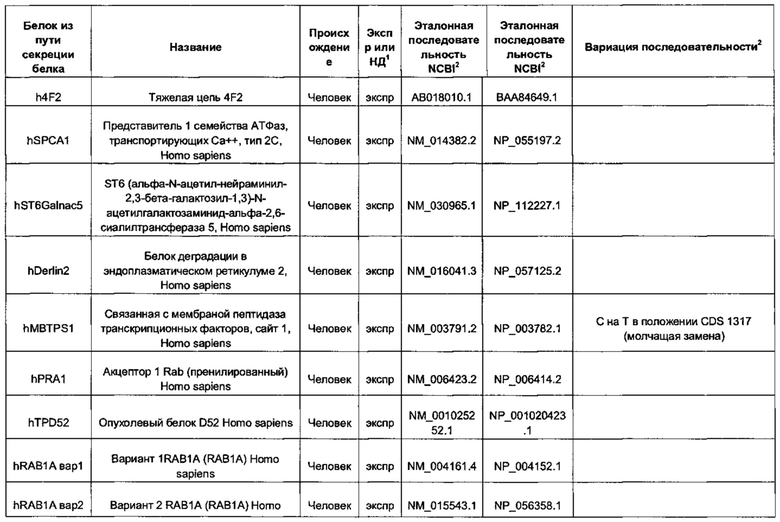
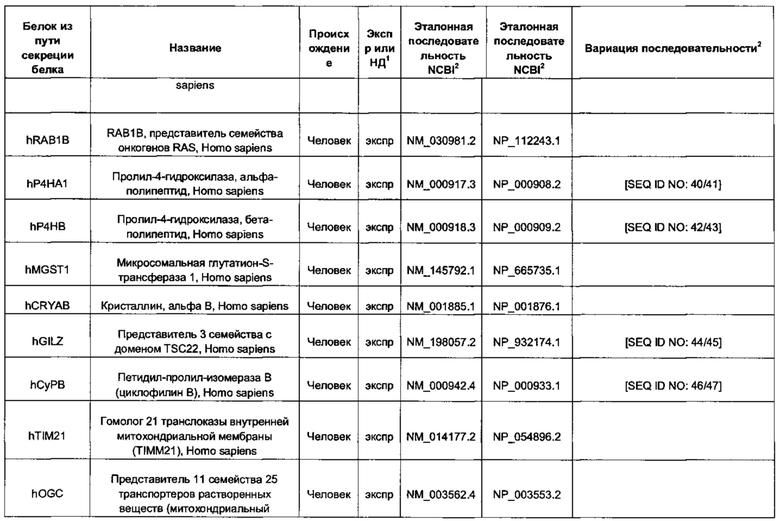
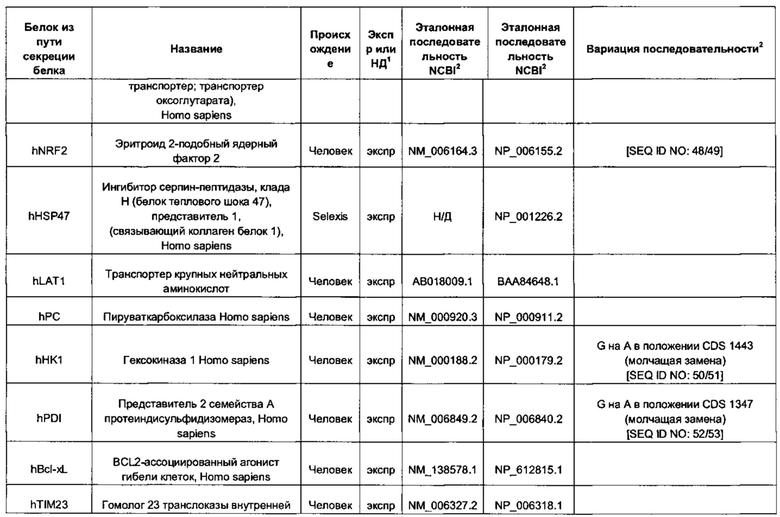
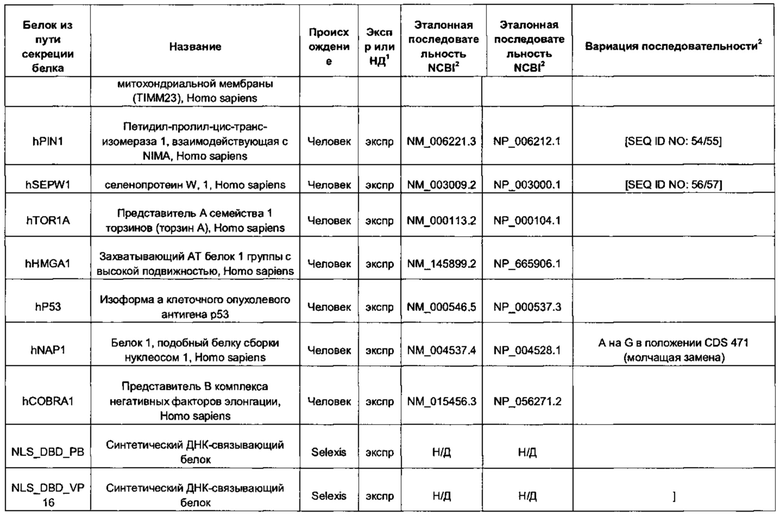
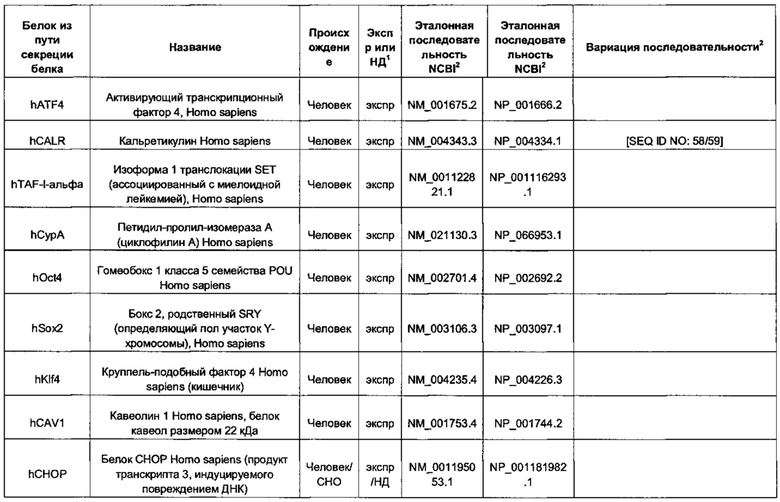
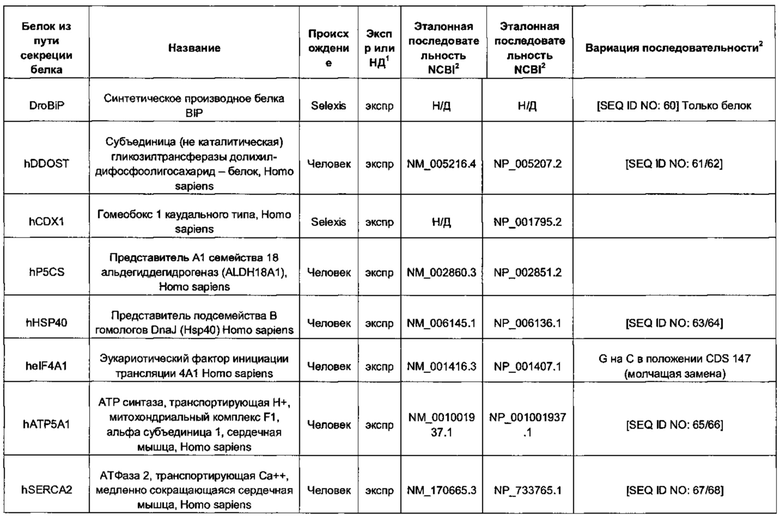
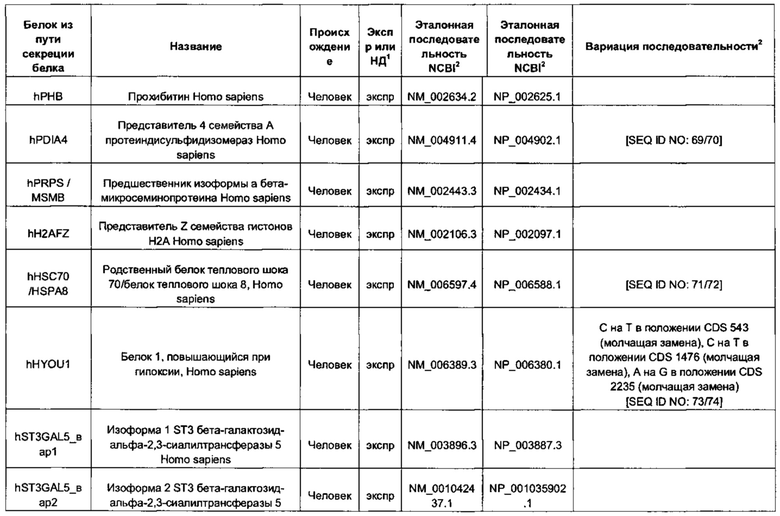
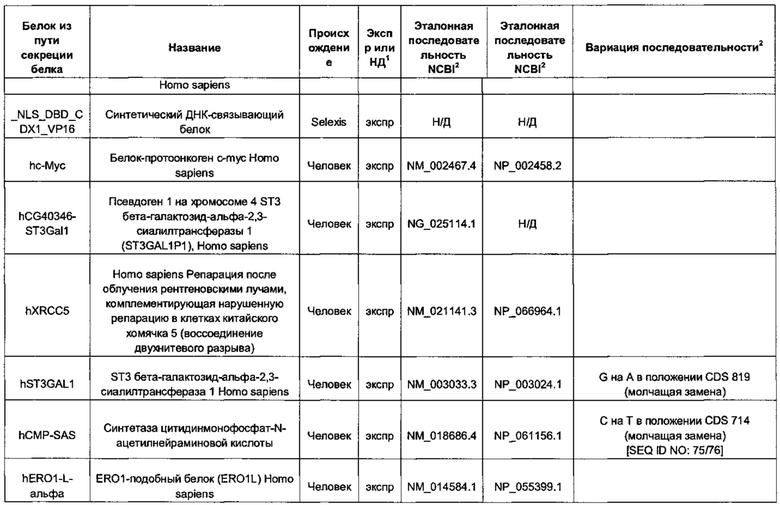
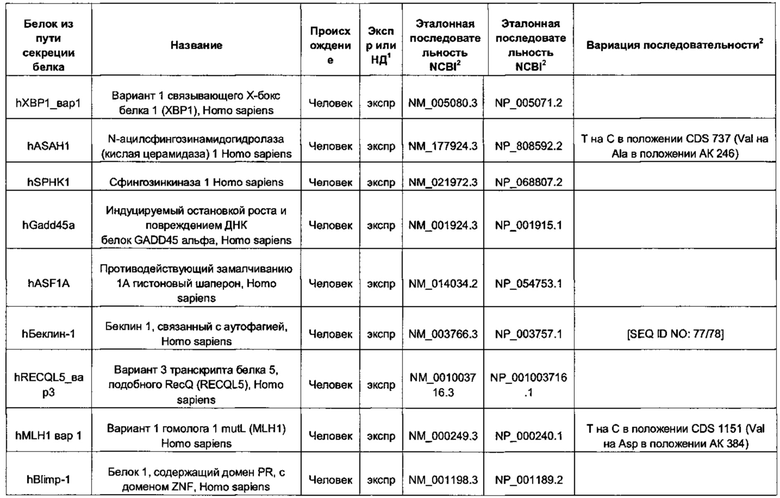
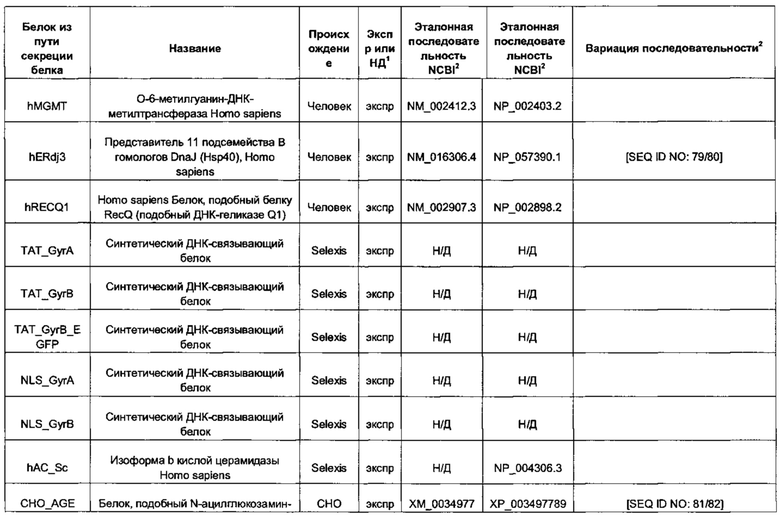
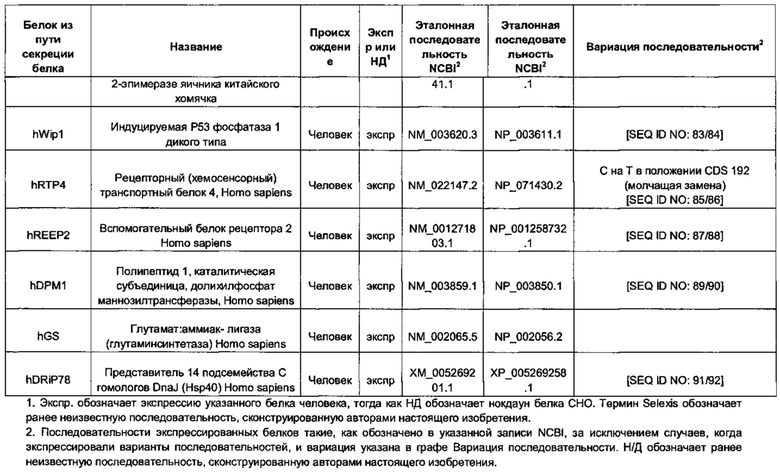
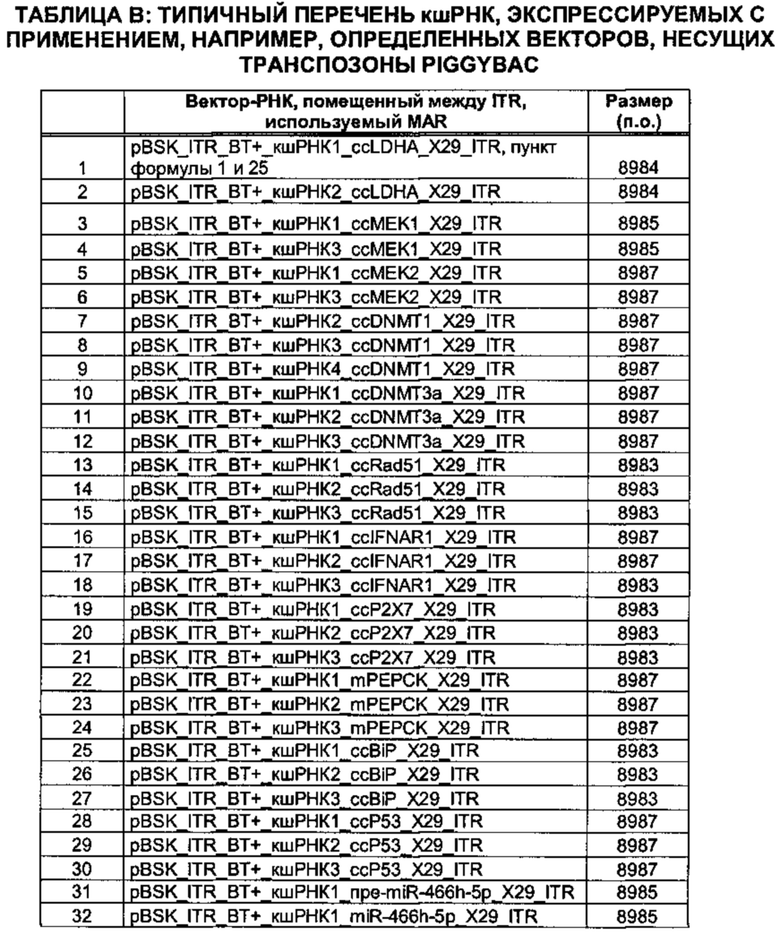


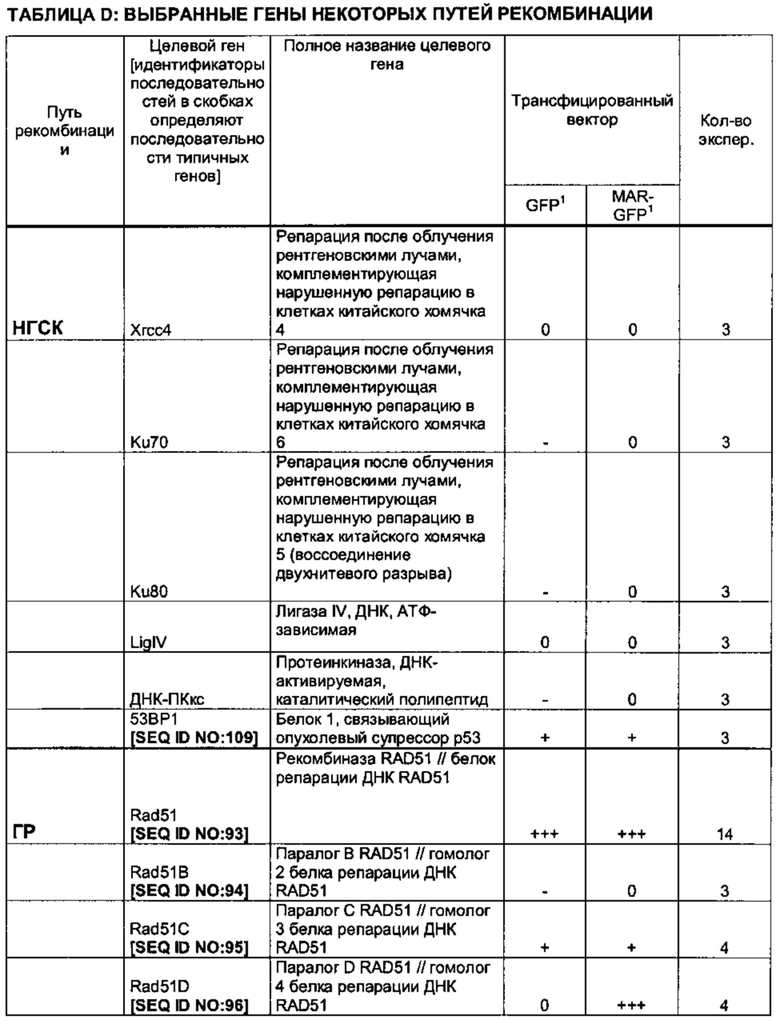
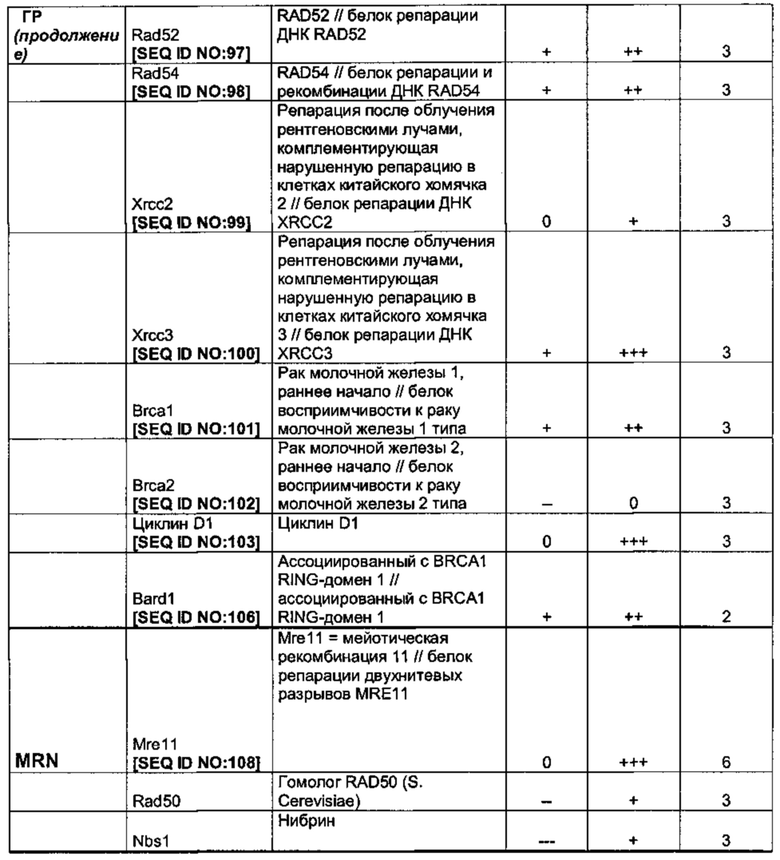
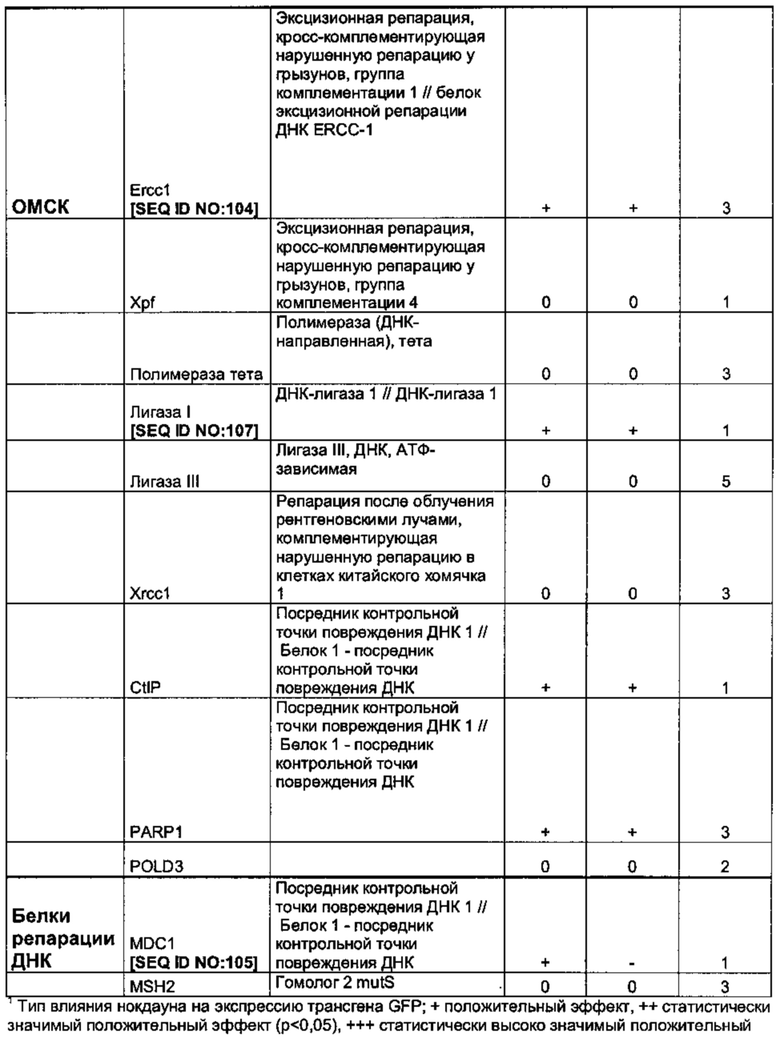

--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> SELEXIS S.A.
<120> УЛУЧШЕННЫЕ ЭКСПРЕССИЯ И ПРОЦЕССИНГ ТРАНСГЕНА
<130> 3024-192PCT
<160> 116
<170> PatentIn версии 3.5
<210> 1
<211> 3616
<212> ДНК
<213> Homo sapiens
<220>
<221> MISC_BINDING
<222> (1)..(320)
<223> MAR 1_68 хромосомы 1
<400> 1
gactctagat tataccaacc tcataaaata agagcatata taaaagcaaa tgctcttatc 60
ttgcagatcc ctgaactgag gaggcaagat cagtttggca gttgaagcag ctggaatctg 120
caattcagag aatctaagaa aagacaaccc tgaagagaga gacccagaaa cctagcagga 180
gtttctccaa acattcaagg ctgagggata aatgttacat gcacagggtg agcctccaga 240
ggcttgtcca ttagcaactg ctacagtttc attatctcag ggatcacaga ttgtgctacc 300
tattgcctac catctgaaaa cagttgcttc ctatatttca tccagtttaa tatttattta 360
aaccaagaag gttaatctgg caccagctat tccgttgtga gtggatgtga aagtaccaat 420
tccattctgt tttactatta actatccttt gccttaatat gtatcagtag gtggcttgtt 480
gctaggaaat attaaatgaa tggcatgttt cataggttgt gtttaaagtt gttttttgag 540
ttaaatcttt ctttaataat actttctgat gtcaaaaaca cttagaagtc atggtgttga 600
acatctatat agggttggat ctaaaatagc ttcttaacct ttcctaacca ctgtttttgt 660
ttgtttgttt ttaactaagc atccagtttg ggaaattctg aattagggga atcataaaag 720
gtttcatttt agctgggcca cataaggaaa gtaagatatc aaattgtaaa aatcgttaag 780
aacttctatc ccatctgaag tgtgggttag gtgcctcttc tctgtgctcc cttaacatcc 840
tattttatct gtatatatat atattcttcc aaatatccat gcatgggaaa aaaaatctga 900
tcataaaaat attttaggct gggagtggtg gctcacgcct gtaatcccag cactttggga 960
ggctgaggtg ggcggatcat gaggtcaaga gatcgagacc atcctgacca atatggtgaa 1020
accccatctc tactaaagat acaaaactat tagctggacg tggtggcacg tgcctgtagt 1080
cccagctact cgggaggctg aggcaggaga acggcttgaa cccaggaggt ggaggttgca 1140
gtgagctgag atcgcgccac tgcactccag cctgggcgac agagcgagac tctgtctcaa 1200
aaaaaaaata tatatatata tatatataca catatatata taaaatatat atatatacac 1260
acatatatat ataaaatata tatatataca cacatatata taaaatatat atatatacac 1320
acatatatat aaaatatata tatacacaca tatatataaa atatatatat acacacatat 1380
atataaaata tatatataca cacatatata taaaatatat atatacacac atatatataa 1440
aatatatata tacacacata tatataaaat atatatatac acacatatat ataaaatata 1500
tatatacaca catatatata aaatatatat atacacacat atatataaaa tatatatata 1560
cacacatata tataaaatat atatatacac acatatataa aatatatata tacacacata 1620
tataaaatat atatatacac atatatataa aatatatata tacacatata tataaaatat 1680
atatacacac atatatataa aatatatata tacacacata tatataaaat atatatatac 1740
acatatatat aaaatatata tatacacata tatataaaat atatatatat acacatatat 1800
ataaaatata tatacacaca tatatataaa gtatatatat acacacatat atataaaata 1860
tatatataca catatatata aaatatatat atacacatat atataaaata tatatataca 1920
catatatata aaaatatata tatatatttt ttaaaatatt ccaattgtct cactttgtgg 1980
atgagaaaaa gaagtagtta gaggtcaagt aacttggcct acatcttttc tcaagattgt 2040
aaactcctag tgagcaataa ccacatcttc attttctttg tataaaacaa gaaagtttag 2100
catgaaaaag gtactcaatt acaaatgtgt tggattgaat tgaagaccct tggaagggga 2160
ttttgtacct gaggatctct ttcttttggc catattgttc aatggacaaa atttagcctt 2220
cgaaggcagg ccgatttgag gttaatacta cctttaccac ttgatagcta tgtgaccttg 2280
gccatgtggt ttcaacagtc tgaacctcat tttctctgtg tatgtgtggt cctccttaca 2340
agtttgtgaa aaatgtgaag tccttagcca tgatagccca atataacagg ctaaatgata 2400
ataggtttat gttcttttcc tttatattct cagataagca ctgtccaagt ttgaggtgtt 2460
ttgaggtctc gcctgatttg gattgtttga gtttatgcta ttctttgaat tctttgagct 2520
gttctgaagc agtgtatcat gaacaaaaac atccccagtt cagtccaaac ccctggttac 2580
atatcattct tatgccatgt tataaccagt ttgagagtgt tccctctgtt attgcattta 2640
agtttcagcc tcacacagaa attcagcagc caatttctaa gccctaagca taaaatctgg 2700
ggtggggggg ggggatggcc tgaagagcag cattatgaat agcaccatta taattaatga 2760
tctctcagga agatttacaa tcacaggtag cagataaaac aaatagtact gcttctgcac 2820
ttcccctcct tttattcgct atgaaatttt atgggaaatc agtccagtga aaaatgtaag 2880
ctcttaatct ttcccagaaa tcctacctca tttgatgaat actttgaggg aatgaattag 2940
agcatttttt tcttttatag tctacttcgc atttacgaag tgaggacggt agcttaggct 3000
gcctggccaa ctgatgagaa ggtcagaggc atttttagag acctctgttg tctttcattc 3060
atgttcattt tccacaaggc aagtaatttc caacaaatca gtgtcttcat tagtaataag 3120
attattaaca acaataatag tcatagtaac tattcagtga gagtccatta tatatcaggc 3180
attctacaag gtactttata tacatctgag taaacctcac acaattctac agggaggtat 3240
ttctatcccc atttaacaaa taaggaaacg aagtccaagt aaattaactt gcccaaggtc 3300
acacagatag tacctggcag aacaggaatt taaacctaaa tttgtccaac tccaaaagca 3360
gccttctatt tgttataaat gctgcctctc attatcacat attttattat taacaacaac 3420
aaacatacca attagcttaa gatacaatac aaccagataa tcatgatgac aacagtaatt 3480
gttatactat tataataaaa tagatgtttt gtatgttact ataatcttga atttgaatag 3540
aaatttgcat ttctgaaagc atgttcctgt catctaatat gattctgtat ctattaaaat 3600
agtactacat ctagag 3616
<210> 2
<211> 4624
<212> ДНК
<213> Homo sapiens
<220>
<221> MISC_BINDING
<222> (1)..(4624)
<223> MAR 1_6 хромосомы 1
<400> 2
ggatcttaaa tctattttat ttatttattt ttcatgtggc caataccctc cacccccttc 60
ttctgtctct ttcaacttat tgtggttacc ttgaggctac ctgagacagt aggcttgggt 120
ggggaagtat gcattctaag tgtaaagttt gatgagcttt gacaaatgtc aacccatgta 180
ccagaacatt ttcatcaccc ataaaatctc ccttgtgtca cttgccagtc agtgtctatt 240
ctagtatcca actcctggct ccaagaaacc attgaactgt tttctgtcac tataaattag 300
atttgtcttt tctagagttt catgtaaatg gaatcataca ctaagtactc tttgtgcctg 360
gcttctgctc agcataatgt ttttgagaat cattcatgct gctgcatgtt ttcagtagtt 420
cattttttta aataggtgaa ttgtaactca ttctgtgaat ataccatatt ctgtcttcca 480
tttatctgtt agtggatctt taggtcgttt ctagttttgg gctattgcaa ataaagctgc 540
tgtaaatatt aatgcacaag ttttccatgt tcatatgttt catttcactt aggaaaatac 600
ctaagagagg aattgcacat attaaaaaaa ttttaaaaac tactaagctg ttctccaaaa 660
tggttgtaca atttttattc ccaagagcaa tatgagtgtt taattgctcc acattctcac 720
caacacttgg tgcttgttag ttttattttc attgttttca ttgttatgtc tgtgaggcag 780
cattgatgtg catgtctctg agtgtcatct tagcggtgat gctgagcatc agttcacgtc 840
cttataggcc gtttgtatat ctgctttgtg aaatgtctgt tcaaatcttt tgcctatttt 900
aaattgagtt gtgttcgtct tcttaggatt aagtaatgag ttaaaaatat ttctgataca 960
aatctttcat tatatatttc taatgctttc tcatctatag tttattttct catattcttt 1020
aactgtatct tttgaagagc aaattttact tttgattatg cccaatttat caagttttta 1080
tatggctctt ttgattatgc ccataatcac attagacttt gcctaaccca agtttgcaga 1140
gattttttct tttatgcttt tatctagaaa ttttgtagtt ttaggtttta aaaaagttta 1200
atttatttat ttgagacagg gtattgctct ttacatatac tggagtgcag tgatgcaatc 1260
atggctcact gcagcctcaa cctcttgggc tcaagcggtt ctcccatctc agagtcctga 1320
gtagctggcc aggtgcatgc cagcttcaat gtgtttttca tttgcatttc cctgataatt 1380
attgacgttg agcatttttt tcatatatca gttagctatt tgtacgtctt cttttgagaa 1440
acatctattc gggtcttttg cccattttaa agtcagatgg tttgtttgtc agctattgag 1500
ttgtttgagt tccttgtata ttctggatat taccatcttg tcagatgcac agtttgcaaa 1560
tttttttttt ctattttgta ggttgtctct ttctctgttg tttcctccgg tatgcagaag 1620
ttttttagtg tgatgtaatt tcatttgtct gtttttgctt ttgttgcctg tactttctta 1680
ttcttatcca aaaaatcttt atctagatca atgtcacgaa gagtttctcc tctgttttct 1740
tcgagtagtt ttttataatt ttgggtatac atttaagtct ttaatctatt tggaattgat 1800
ttttgcatat ggtgagagat cagagtctaa tttcatactt ttggatgtgg aaagctagtt 1860
ttttcagcac catttattga agagactgtc tcttctccaa tgtgtgttct ttgtgccttc 1920
gtcaaaaatc agttggctgt gcgtggattt atttctgtgt tctctatttt gttccattgg 1980
tctagtttta gccttaaatt taggtctgca attttttttt ttttgtatat ggtgtgaagt 2040
aagagtcaaa gttcattatt tttcatatgg atatgtaatt actccagtac catcatttag 2100
tttgaatgga ctgtcctttc tccatggaat tacatgggca tcttttgtct gaaaccaatt 2160
atgtatgttt acgtatgtgt atgtttatgc atatgttata ggtttaatat atattaatat 2220
atataatata taatatataa atattaatat gtattatata atatatatta atatattata 2280
ttatattact atataaataa tattaatata ttatattaaa atattaataa atatatcata 2340
ttaaatatta tattaattaa atattaataa atatattata ttaatatatt tatatattaa 2400
acctataaca tatgcatata cttatttata tataacatgc atgtacttat ttatatatac 2460
aatatatatt tatatattat ataatatatt atatgtattt atatattata tatcatatat 2520
tatatgtatt tatatattat atatcatata atatatatat ttatattata tatattatat 2580
gatatataat attatataat gtattaatat atattaaacc tatatttata attctggact 2640
cactattttg tttcattggt gtctgtgtgt atctaaccct atgccaataa tgtactatct 2700
taattaccat agctttatag taagctttga aatcagatag tgtatttttt atcattgttt 2760
tttaaaataa tagtttatct ttttatttga atttgtaatc agctagtcag tttctgcaaa 2820
aagcttactg ggattttgct tggaattatg ttacatctgt agcatgtact atccaatatt 2880
ctagccttta tccacatgtg gctattaagg tttaaattaa ttaaattaaa atttaattaa 2940
ttaaaattaa aacttaataa ttggttcctc attcacacta ccatatgtca agtgttcaat 3000
agccacatat ggtcaatgtc ttggaaaagt caatacagta catttccatt attgcagtaa 3060
gttctgtcaa acagcactat cgtagaccga ttaggagaga actgacttaa cagtattgga 3120
tgctccagtc aatgaacatc tttttttttt tcatttattt cagtagtctc tgcagtatat 3180
tatagatttc agtttacata ttttgcatat attttattaa atgtataacg gtagaagtac 3240
tattattgga tgatgtgttc tatagatgta ttttaggtca agtttgttga tagtgttgtt 3300
taaatctcgt atacctcttg atttttttat ttacttgttc tttgaattac tgagacagga 3360
atgttatatc cttaactata tttgtgaatt tattcacttc ttccttcagt tctgttaact 3420
tttgcttagg tgctttttaa aaatgaaact ttcaatctct gccttttaat tgtagcattt 3480
agaccattta cattcaatgt aattatcaat atcagtttat ttaagtctga agttgtgcaa 3540
tttttcctct acctatatta taaatctttc tatatacaaa acacatgcta tgttttctgc 3600
atatgtttta aatgacaccc ggaaagcatt gacactattt ttgctttagg ttatctttca 3660
aagatgttaa aaatgagaaa gaaatattct gcatttatcc atacacttat tatttgcaaa 3720
ggttttttta aatacctttg tgtagatttc agttaccaac ttgtatttcc ttcagcttga 3780
agaacttaca atttcttgta ggacaggtct ctgacaacaa attatctcag cttttctttg 3840
tctaaaaaag ttattgcctt tatttttaaa atatattttc actggatatt gaattttagg 3900
tgataatctt tttttttttg ttagcacttt aaatatgtct tctaatgtcc tcttgctttc 3960
atagtttctg atgagaagtc tactgttatt agtatctctt tgtgtgtgtc tctctttttt 4020
ccctctctgc tattatggct attttttttt tttttttttt ttttggtcac tggtgtcagc 4080
aatttaatta tggtgtgcct tggtatgttt tgtgtgtgtg tgtgtgtgtg tgtgtgtgtg 4140
tgtgtgtgtg tagctgatgt tctttgagct ttagaatctg tgagtttgta gttttcatca 4200
attatttttt cttttcattc cttttattta ctcatgttcg tgttttattt tatattttta 4260
agaattttgt gcgtatttgt aataactgtt taaatgtcat ttgtgaattc cattgcttct 4320
aggtaggatt ctattgacag atattttttc cctgacgaga ggtcatactt tccttattct 4380
tcatgtatct agtggttttt ggttgaatac tggatatttt gaattttatg ggagtgctga 4440
attctacaat attccttaaa aatgtgttgg attttgtttt agcagatagc tatcttactt 4500
gaagatcaat ttcatatttt ttgatgttca ttttttcatt tattaaagaa taggtccatg 4560
gtagagttta ctgatatcaa cctttctggt gtctctaata aatgcaacat attcaataag 4620
atcc 4624
<210> 3
<211> 3354
<212> ДНК
<213> Homo sapiens
<220>
<221> MISC_BINDING
<222> (1)..(3354)
<223> MAR X_S29 хромосомы X
<400> 3
gatcccttta taaaaccaca atataatgga gtgctataat ttcaaacagt gtttggtctg 60
ctggcagagt ggtcattcta acagcagtca cagtagagta gaaataagac tgcagtatat 120
ctaaggcaaa aagctgaggt ttcaggagct tgaaggtaaa gaggaagaaa gaaatgggaa 180
tgggaattgg aaagacaaat atcgttaaga gaaaattgct tttaggagag gggaaagaat 240
ctatgtgtac ttaagactat ggaatcaatc ccatttaagc tgggaaacta gtttcatata 300
taactaataa attttattta cagaatatct atttacctga tctaggcttc aagccaaagg 360
gactgtgtga aaaaccatca gttctgtcat attcctaaaa aaaaattaaa aagttaaaaa 420
taaataaata ataaaacttc ttttctttca aaataatcaa ggtgcttatt cacatccatt 480
ccaatttggg gaaatactta ttttcctatg attagcgaag agaaaagtaa cttgcatttc 540
aattcaagtt gatacatgtc acttttaaga ggtcaactaa tatttgctag ttgagctaac 600
catataggct ttaaatactt tcatagtaga aagaaaatga aaatcattag tgaactgtat 660
aaaatagatc atactttttg aaagaatcag actgaagttt ccgaaaaaaa gaagtaagct 720
tcaatgaaaa ggtaagtgaa tttagcattt actcagcatc tactatggac ttaacaccta 780
acagtagata atctgaaggc aaacatattt gtatagggac tgcagaatga tagatgataa 840
atatcatctc ttctatttga atgaatattt tttcaaatct ttcacacaca gtggtttgct 900
atggaaagat ttgtagtaca ttaaacaaat ctgaagatgg agttagaaag cttaggctat 960
gttttgagca caacatataa tttctctgtg attgtttctt catctttcaa atgaggttac 1020
tgtgaagatt aaatgagata actaaatgat gataaaataa tgtaatctta gcagcacctt 1080
atttaatctg tgcaacaact ctgtgaagtg agtagggctc agcttcagtc acttctctgc 1140
catttattaa ctaagatagt ttggaaagtt acccatctct tcagctgtaa aatgatgagg 1200
atcataccta ttttatgggg ctgcttttag gtacaaatat acaggcaagc actttgttaa 1260
tactaaagca ttacaccaat tagttttact cttttccatt cacacatgaa attaatgtaa 1320
tcagaattct gtagattacc taaatcttct gttaacacgt gatatgcagt tcaggttaaa 1380
tgtcagttga gttaccaaag cacatacata ctcaccaccc tatccaaatc tacaagcctc 1440
ccagtttgtc ttcactattt tggttaaatt aatatgaatt cctagatgaa aatttcactg 1500
atccaaatga aataaaaaat atattacaaa actcacacct gtaatctcaa cattttggga 1560
ggccaaggca ggtagatcac ttgaggccag gagttcaaga ccagcctgat caacatggtg 1620
aaaccctgtc tctactaaaa atacaaaaat tagccaggtg tggtggcatg tgcctgtagt 1680
cctacctact cgggaggctg aggcacaaga atcgcttgaa tgtgggaggt ggaggttgca 1740
gtgacctgag atcgtgccac tgcactccag cctaggcaac agagtgagat catgtgtcat 1800
atatatatat atatatatat atatatatat atatatatac acacacacac acatatatat 1860
atacacatat atatacgtat atatatatat gtatatatat acatatatat acatatatat 1920
atatacgtat atatatacgt atatatatat caatgtaaat tatttgggaa atttggtatg 1980
aatagtcttc cctgtgaaca cagatcataa aatcatatat caagcagaca aataagtagt 2040
agtcacttat atgcttatac ttgtaactta aagtaaaaga attacaaaag catatgacaa 2100
agactaattt taagatatcc taatttaaat tgttttctaa aagtgtgtat accattttac 2160
ctatcatatg aataatttag aaacatgttt ataaaattaa tgtccaaatc cattcaaaag 2220
ttttgtaatg cagatcaccc acaacaacaa agaatcctag cctattaaaa aagcaacacc 2280
acctacatat aatgaaatat tagcagcatc tatgtaacca aagttacaca gtgaatttgg 2340
gccatccaac actttgagca aagtgttgaa ttcatcaaat gaatgtgtaa tcatttactt 2400
actaatgcca atacacttta aggtaatctt aagtagaaga gatagagttt agaatttttt 2460
aaatttatct cttgttgtaa agcaatagac ttgaataaat aaattagaag aatcagtcat 2520
tcaagccacc agagtatttg atcgagattt cacaaactct aactttctga tacccattct 2580
cccaaaaacg tgtaacctcc tgtcgatagg aacaacccac tgcagggatg tttctcgtgg 2640
aaaaaggaaa tttcttttgc attggtttca gacctaactg gttacaagaa aaaccaaagg 2700
ccattgcaca atgctgaagt acttttttca aatttaaaat ttgaaagttg ttcttaaaat 2760
ctatcattta ttttaaaata cggatgaatg agaaagcata gatttgataa agtgaattct 2820
tttctgcaat ctacagacac ttccaaaaat cactacagac actacagaca ctacagaaaa 2880
tcataaataa acaagtgcta gtatcaatat ttttaccaaa aaatggcatt cttagaattt 2940
tttataggct agaaggtttg tacaaactaa tctgccacgg attttaaaat atgagtgaat 3000
aaattatatt gcaaaaaaaa tcaggttaca gagaactggc aaggaagact cttatgtaaa 3060
acacagaaaa catacaaaac gtatttttaa gacaaataaa aacagaactt gtacctcaga 3120
tgatactgga gattgtgttg acatattagc attatcactg tcttgctaaa acataaaaat 3180
aaaaagatgg aagatgaaat tacaatacaa atgatgattt aaacatataa aaggaaaata 3240
aaaattgttc tgaccaacta ctaaaggaag acctactaaa gatatgccat ccagcacatt 3300
gccactctac atgtggtctg taaaccagca gcatagggat cctctagcta gagt 3354
<210> 4
<211> 5463
<212> ДНК
<213> Mus musculus
<220>
<221> MISC_FEATURE
<223> Конструкция S4_1-703_2328-5457 с целыми сайтами AvaI
<400> 4
ctcgaggtct caagataaga atgactgctg taactcaaat ccaccaaagc tatttgtgtt 60
agaatgcttt cctttggtaa taacataata ccacagagtg agtgaatgta tcaagcaaag 120
tactcactca taatctctcc acccaaatga ctttgtcttc taaaattaaa cccttcccag 180
aggcctctcc ccttaatacc atattgggct cttcacactt cttccaacat cgccttccat 240
cctggccctt ccaacctccc ttctgtttgt gctaggaaca gctcaaggcc tcctatctac 300
cacagagtta catggcttgc cccttgccaa ccccccagta ccacacagtg agtgcaaaat 360
ctcaccacat tcagaaccca gtcactattc aaatcatatt ttaacctttg cagtactgac 420
tacttttgat tcatctaaac attactgaac tttattctag aaaacattta agaaatttgt 480
agttaggttc atcctttgag accttacatt taatttcttt ctatgtaaac ggaaagcatt 540
gttcagtccc acgctcatta tggcaaccca cttccaagta cttcgtttac tacgtgggct 600
ggaatcatac agttttctgt tgtgcttgtg ggagcagatc cccctaacct ctgctgattt 660
ttctcaccac ttatcataca tttattacat gcatgcactg ctgtgtgagt ttctaaatac 720
ttgggtagca attctctact attactttaa ttttcctact tgtctgcaaa tacgaaaagt 780
agcttgaaag aacttcagat ctttgttgtt atctgttgca aacactccat ttttctgttg 840
tagcaaaaaa aaaaaaaaag acatccatag ttgtcaatga gaatgcaaga tacatacatt 900
ctgcacctgt gtgctaacat aagtggctgc cctgtgactc agagattgct tgtccttctc 960
ctaagcctat ccttttttgt tactttggat acttttgttc aatgaatcca gaaaaagtgt 1020
ttttcagatt caccatgtga ccctcattta aaacctgtaa tccccctatg gttaagttcc 1080
tgcttttgtt tctgttttct ttctttcagt aaaaggaatt gaacccagtc cttccactta 1140
ctatctgagc atatggctct tttagattat gatgttggtg gtgttcattg gtctcaccaa 1200
aatgctaaag aagccttcat cttctacttg tgggtagtct ttacattcat tactgcaagt 1260
ttagtttatg tggtagtacc agatcctttg cttcttttga cttcatgcct acctaacagc 1320
agctctttcc tttagttaag cttatgaaat agtgtttctc tcatgtttcc tctatattct 1380
ctcttttgcc ttcctgtttc ttcctgttga ttccatccca ttggagtgaa atcttatgat 1440
cttttggcat caacaaagtg atctgcatcc aaataattcc acatctcatt ccatgttgac 1500
tgtggatcta tatatatata tatgtatata tgtatatatg tatatatgta tatatgtata 1560
tatgtatata tgtatatatg tatatatgta tatatgtata tatgtatata tgtatatatg 1620
tatatatgta tatatgtata tatgtatata tgtatatatg tatatatgta tatatgtata 1680
tatgtatata tgtatatatg tatatatgta tatatgtata tatgtatata tgtatatatg 1740
tatatatgta tatatgtata tatgtatata tgtatatatg tatatacgta tatatgcata 1800
tacgtatata tgtatatatg tatatatgta tatatgtata tatgtatata tgtatatatg 1860
tatatatgta tatatgtatg tatgtatgta tgtatgtata tatgtatata tgtatgtatg 1920
tatgtatgta tgtatgtatg tatatatgta tatatatatg tatgtatgta tgtatgtatg 1980
tatgtatatg tgtatatgtg tatatgtgta tatgtgtata tgtgtatata tgtatatatg 2040
tatatatgta tatatgtata tgtgtatatg tgtatatgtg tatatatgta tatatgtata 2100
tatgtatata tgtatatata taacatagta ttaaattata tatacatata taagtgaaat 2160
gtcacaatct tctagaactt gctctgtatg tccacttaac atggtagagt gagctatgtc 2220
agcattttct atttcctgtg aatcattctg tgtgttgcca agaagaaata tgatatattc 2280
tgaggttatg aaatgatatt ttggtcatca tgtttctcat cctattttca tattacctaa 2340
atacttttgc ttttaaaatt attattatta ataataatat aattatttat acaataatat 2400
ttaaataata tatttattta atataattat tatatttcac ataaaagcaa tagttccagt 2460
gttacaaatt gtaggcaact gggctgttct gattatctaa gttgggccca ggatatgtgc 2520
tgaatagtta aagcacatgc ccagcatgta tgagggtaaa aggatgggtg gatgtagtga 2580
cccatttgta atttaagcct tagcaggcag aggtgtgacc catagtgcaa agtacatagt 2640
cattataagg tcatctatat cacaatctct ggattagatt gattgaacct gctcagtgac 2700
caatgtgtta gcaatataca ggaggatgat aacatcaacg tcagaagaca cattgaaggg 2760
cttacaaata gtgcccattt actttaatac agaaaaattc aatgtaccct ctaggcaatt 2820
tcaactttta gtctcttggt aggatagtct acatttagaa tggctaattc ataaattaga 2880
aagcttcttc accccctact tttctggtta tttctctatg aatgtggtag gcatgagtta 2940
gtacacatgt ttccatgtac atgtgtttct atgtgtctgc atgcatatgg tagaatgtac 3000
tcatattcta tgtacagtta gaacaatatt tatattgtca aagaaatcaa aaggagtatt 3060
ataagcttca gaaataagga taagtttgaa atattcattg ttttattttt tacagtattt 3120
tttcctttga gaattctatg taaagtactt tgaacatatt tgccttcaac tcctccctca 3180
ctttcaccct ctcttcattc ctccctttcc tttccactca aagttgagat tcctttattt 3240
atttatttat ccttcaaata tcactggtac tatccacatg atctcaggat tgaggtctgc 3300
tctgacgtgt catcctgctt tcatgcaatg gccttatagg tggaacaaca ttatgaacta 3360
accagtaccc cggagctctt gactctagct gcatatatat caaaagatgg cctagtcggc 3420
catcactgga aagagaggct cattggactt gcaaacttta tatgccccag tacaggggaa 3480
caccagggcc aaaaaggggg agtgggtggg caggggagtg ggggtgggtg gatatggggg 3540
acttttggta tagcattgga aatgtaaatg agttaaatac ctaataaaaa atggaaaaaa 3600
aaagtttcta atgtgtgttt ctagaaactt cctctcttaa agcaacaaca tgtccatgag 3660
caatatagaa ttgaagatca ccatcaaatc ctctttattc ctcattgttt ccatcatgta 3720
ctaccagacc tctttaaagt gtagtacagt gtgttaggaa atgagcagat tatcctgggt 3780
atgtgctaaa ttagctactg agtcaaaata cattttttgc tgaacattaa gtgtttggtc 3840
atttctgggc aaaagaaaga aagaaagaaa gaaaagaaag aaagaaagga aggaaggaag 3900
gaaggaagga aggaaggaag gaaagaagga aggaaagaaa aaatggatgt aaattgttct 3960
gacagcatct gtctgagtca ggcagtggaa tgaaggagga atcctagaga atgcacagga 4020
aagcagccca aggagagtgt gggctgaaag gcatcatgtt agaaacatgc actcgatgac 4080
agaaccttga gaaaaaggaa ctcaagcaaa agcacttatt taaaattgta aaacgcactt 4140
tattcatagc catgggggat gtcaatattc caagcataag aatgatcagt ttccaatcac 4200
tgtgaacccc caaaacacaa agtgaaaacc cactacttta tttgatgaga tttggggttg 4260
ctctattaat ttataaaatc agagtaagac acgatataaa tgaaacgatt gtagttctaa 4320
agcagcggca cttccctgaa cagtgtcatt ttgacaagta actgctaaca tcttcaggtc 4380
acagcgactg aagaaaaagt agggaaagaa ggctggctgt gctgtttgac attttctttt 4440
cttatctggt gacatgaaga gaagctctgg gtccccctac tcttgttcat atatctgttg 4500
cttttatgct gcatcctgag gtttgaagaa atgcatttgg cactgagaaa agatgaggag 4560
agaatgcctt ggacatggtc ctaacatgct ttggtactga gaaaagagag cagaggagat 4620
gacatagaat aggagagata atttggccta ttttggcctt catctgagtg atagatttta 4680
cttaacaaat agaaacaaag ttttacttat aaacagaacc aatgacctgt gtcatctctg 4740
atatattgag ctttgaattc agtgaaatta tgaactaaat atatcactcc ataattttct 4800
aagagggcta tttgtatagt ttcagtgata gtgtgacaaa gtgtaatcta aatttctaaa 4860
aagtaaaata agtagataaa atagtaggta gaatagtata ataatagaat aagtataggt 4920
atggactaga ataaatagac aaaatagtag ataaaatgct aatgattttg ttgacagggt 4980
aatcatgaat atttttatta tttagctaaa gaaccaatgt tcatgtactc aagaagtgta 5040
ttgaggaact taggaaatta gtctgaacag gtgagagggt gcgccagaga acctgacagc 5100
ttctggaaca ggcggaagca cagaggcact gaggcagcac cctgtgtggg ccggggacag 5160
ccggccacct tccggaccgg aggacaggtg cccgcccggc tggggaggcg acctaagcca 5220
cagcagcagc ggtcgccatc ttggtccggg acccgccgaa cttaggaaat tagtctgaac 5280
aggtgagagg gtgcgccaga gaacctgaca gcttctggaa caggcagaag cacagaggcg 5340
ctgaggcagc accctgtgtg ggccggggac agccggccac cttccggacc ggaggacagg 5400
tgcccacccg gctggggagg cggcctaagc cacagcagca gcggtcgcca tcttggtccc 5460
ggg 5463
<210> 5
<211> 2957
<212> ДНК
<213> Лизоцим цыпленка
<400> 5
tctagaaaac aatatatttc caaatgaaaa aaaaatctga taaaaagttg actttaaaaa 60
agtatcaata aatgtatgca tttctcacta gccttaaact ctgcatgaag tgtttgatga 120
gcagatgaag acaacatcat ttctagtttc agaaataata acagcatcaa aaccgcagct 180
gtaactccac tgagctcacg ttaagttttg atgtgtgaat atctgacaga actgacataa 240
tgagcactgc aaggatatca gacaagtcaa aatgaagaca gacaaaagta ttttttaata 300
taaaaatggt ctttatttct tcaatacaag gtaaactact attgcagttt aagaccaaca 360
caaaagttgg acagcaaatt gcttaacagt ctcctaaagg ctgaaaaaaa ggaacccatg 420
aaagctaaaa gttatgcagt atttcaagta taacatctaa aaatgatgaa acgatcccta 480
aaggtagaga ttaactaagt acttctgctg aaaatgtatt aaaatccgca gttgctagga 540
taccatctta ccttgttgag aaatacaggt ctccggcaac gcaacattca gcagactctt 600
tggcctgctg gaatcaggaa actgcttact atatacacat ataaatcctt tggagttggg 660
cattctgaga gacatccatt tcctgacatt ttgcagtgca actctgcatt ccaactcaga 720
caagctccca tgctgtattt caaagccatt tcttgaatag tttacccaga catccttgtg 780
caaattggga atgaggaaat gcaatggtac aggaagacaa tacagcctta tgtttagaaa 840
gtcagcagcg ctggtaatct tcataaaaat gtaactgttt tccaaatagg aatgtatttc 900
acttgtaaaa cacctggtcc tttttatatt actttttttt ttttttaagg acacctgcac 960
taatttgcaa tcacttgtat ttataaaagc acacgcactc ctcattttct tacatttgaa 1020
gatcagcaga atgtctcttt cataatgtaa taatcatatg cacagtttaa aatattttct 1080
attacaaaat acagtacaca agagggtgag gccaaagtct attacttgaa tatattccaa 1140
agtgtcagca ctgggggtgt aaaattacat tacatggtat gaataggcgg aattctttta 1200
caactgaaat gctcgatttc attgggatca aaggtaagta ctgtttacta tcttcaagag 1260
acttcaatca agtcggtgta tttccaaaga agcttaaaag attgaagcac agacacaggc 1320
cacaccagag cctacacctg ctgcaataag tggtgctata gaaaggattc aggaactaac 1380
aagtgcataa tttacaaata gagatgcttt atcatacttt gcccaacatg ggaaaaaaga 1440
catcccatga gaatatccaa ctgaggaact tctctgtttc atagtaactc atctactact 1500
gctaagatgg tttgaaaagt acccagcagg tgagatatgt tcgggaggtg gctgtgtggc 1560
agcgtgtccc aacacgacac aaagcacccc acccctatct gcaatgctca ctgcaaggca 1620
gtgccgtaaa cagctgcaac aggcatcact tctgcataaa tgctgtgact cgttagcatg 1680
ctgcaactgt gtttaaaacc tatgcactcc gttaccaaaa taatttaagt cccaaataaa 1740
tccatgcagc ttgcttccta tgccaacata ttttagaaag tattcattct tctttaagaa 1800
tatgcacgtg gatctacact tcctgggatc tgaagcgatt tatacctcag ttgcagaagc 1860
agtttagtgt cctggatctg ggaaggcagc agcaaacgtg cccgttttac atttgaaccc 1920
atgtgacaac ccgccttact gagcatcgct ctaggaaatt taaggctgta tccttacaac 1980
acaagaacca acgacagact gcatataaaa ttctataaat aaaaatagga gtgaagtctg 2040
tttgacctgt acacacagag catagagata aaaaaaaaag gaaatcagga attacgtatt 2100
tctataaatg ccatatattt ttactagaaa cacagatgac aagtatatac aacatgtaaa 2160
tccgaagtta tcaacatgtt aactaggaaa acatttacaa gcatttgggt atgcaactag 2220
atcatcaggt aaaaaatccc attagaaaaa tctaagcctc gccagtttca aaggaaaaaa 2280
accagagaac gctcactact tcaaaggaaa aaaaataaag catcaagctg gcctaaactt 2340
aataaggtat ctcatgtaac aacagctatc caagctttca agccacacta taaataaaaa 2400
cctcaagttc cgatcaacgt tttccataat gcaatcagaa ccaaaggcat tggcacagaa 2460
agcaaaaagg gaatgaaaga aaagggctgt acagtttcca aaaggttctt cttttgaaga 2520
aatgtttctg acctgtcaaa acatacagtc cagtagaaat tttactaaga aaaaagaaca 2580
ccttacttaa aaaaaaaaaa caacaaaaaa aacaggcaaa aaaacctctc ctgtcactga 2640
gctgccacca cccaaccacc acctgctgtg ggctttgtct cccaagacaa aggacacaca 2700
gccttatcca atattcaaca ttacttataa aaacgctgat cagaagaaat accaagtatt 2760
tcctcagaga ctgttatatc ctttcatcgg caacaagaga tgaaatacaa cagagtgaat 2820
atcaaagaag gcggcaggag ccaccgtggc accatcaccg ggcagtgcag tgcccaactg 2880
ccgttttctg agcacgcata ggaagccgtc agtcacatgt aataaaccaa aacctggtac 2940
agttatatta tggatcc 2957
<210> 6
<211> 3659
<212> ДНК
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<223> 1_68R(заполненный NcoI+XbaI)
<400> 6
gtacccccaa aagaaagaga tcctcaggta caaaatcccc ttccaagggt cttcaattca 60
atccaacaca tttgtaattg agtacctttt tcatgctaaa ctttcttgtt ttatacaaag 120
aaaatgaaga tgtggttatt gctcactagg agtttacaat cttgagaaaa gatgtaggcc 180
aagttacttg acctctaact acttcttttt ctcatccaca aagtgagaca attggaatat 240
tttaaaaaat atatatatat atttttatat atatgtgtat atatatattt tatatatatg 300
tgtatatata tattttatat atatgtgtat atatatattt tatatatatg tgtgtatata 360
tatactttat atatatgtgt gtatatatat tttatatata tgtgtatata tatatatttt 420
atatatatgt gtatatatat attttatata tatgtgtata tatatatttt atatatatgt 480
gtgtatatat atattttata tatatgtgtg tatatatatt ttatatatat gtgtatatat 540
atattttata tatatgtgta tatatatatt ttatatatgt gtgtatatat atattttata 600
tatgtgtgta tatatatatt ttatatatat gtgtgtatat atatatttta tatatatgtg 660
tgtatatata tattttatat atatgtgtgt atatatatat tttatatata tgtgtgtata 720
tatatatttt atatatatgt gtgtatatat atattttata tatatgtgtg tatatatata 780
ttttatatat atgtgtgtat atatatattt tatatatatg tgtgtatata tatattttat 840
atatatgtgt gtatatatat attttatata tatgtgtgta tatatatata ttttatatat 900
atgtgtgtat atatatatat tttatatata tatgtgtgta tatatatata ttttatatat 960
atatgtgtat atatatatat atatatattt tttttttgag acagagtctc gctctgtcgc 1020
ccaggctgga gtgcagtggc gcgatctcag ctcactgcaa cctccacctc ctgggttcaa 1080
gccgttctcc tgcctcagcc tcccgagtag ctgggactac aggcacgtgc caccacgtcc 1140
agctaatagt tttgtatctt tagtagagat ggggtttcac catattggtc aggatggtct 1200
cgatctcttg acctcatgat ccgcccacct cagcctccca aagtgctggg attacaggcg 1260
tgagccacca ctcccagcct aaaatatttt tatgatcaga ttttttttcc catgcatgga 1320
tatttggaag aatatatata tatacagata aaataggatg ttaagggagc acagagaaga 1380
ggcacctaac ccacacttca gatgggatag aagttcttaa cgatttttac aatttgatat 1440
cttactttcc ttatgtggcc cagctaaaat gaaacctttt atgattcccc taattcagaa 1500
tttcccaaac tggatgctta gttaaaaaca aacaaacaaa aacagtggtt aggaaaggtt 1560
aagaagctat tttagatcca accctatata gatgttcaac accatgactt ctaagtgttt 1620
ttgacatcag aaagtattat taaagaaaga tttaactcaa aaaacaactt taaacacaac 1680
ctatgaaaca tgccattcat ttaatatttc ctagcaacaa gccacctact gatacatatt 1740
aaggcaaagg atagttaata gtaaaacaga atggaattgg tactttcaca tccactcaca 1800
acggaatagc tggtgccaga ttaaccttct tggtttaaat aaatattaaa ctggatgaaa 1860
tataggaagc aactgttttc agatggtagg caataggtag cacaatctgt gatccctgag 1920
ataatgaaac tgtagcagtt gctaatggac aagcctctgg aggctcaccc tgtgcatgta 1980
acatttatcc ctcagccttg aatgtttgga gaaactcctg ctaggtttct gggtctctct 2040
cttcagggtt gtcttttctt agattctctg aattgcagat tccagctgct tcaactgcca 2100
aactgatctt gcctcctcag ttcagggatc tgcaagataa gagcatttgc ttttatatat 2160
gctcttattt tatgaggttg gtataatcta gctagagtcg agatctttgg ccatattgtt 2220
caatggacaa aatttagcct tcgaaggcag gccgatttga ggttaatact acctttacca 2280
cttgatagct atgtgacctt ggccatgtgg tttcaacagt ctgaacctca ttttctctgt 2340
gtatgtgtgg tcctccttac aagtttgtga aaaatgtgaa gtccttagcc atgatagccc 2400
aatataacag gctaaatgat aataggttta tgttcttttc ctttatattc tcagataagc 2460
actgtccaag tttgaggtgt tttgaggtct cgcctgattt ggattgtttg agtttatgct 2520
attctttgaa ttctttgagc tgttctgaag cagtgtatca tgaacaaaaa catccccagt 2580
tcagtccaaa cccctggtta catatcattc ttatgccatg ttataaccag tttgagagtg 2640
ttccctctgt tattgcattt aagtttcagc ctcacacaga aattcagcag ccaatttcta 2700
agccctaagc ataaaatctg gggtgggggg gggggatggc ctgaagagca gcattatgaa 2760
tagcaccatt ataattaatg atctctcagg aagatttaca atcacaggta gcagataaaa 2820
caaatagtac tgcttctgca cttcccctcc ttttattcgc tatgaaattt tatgggaaat 2880
cagtccagtg aaaaatgtaa gctcttaatc tttcccagaa atcctacctc atttgatgaa 2940
tactttgagg gaatgaatta gagcattttt ttcttttata gtctacttcg catttacgaa 3000
gtgaggacgg tagcttaggc tgcctggcca actgatgaga aggtcagagg catttttaga 3060
gacctctgtt gtctttcatt catgttcatt ttccacaagg caagtaattt ccaacaaatc 3120
agtgtcttca ttagtaataa gattattaac aacaataata gtcatagtaa ctattcagtg 3180
agagtccatt atatatcagg cattctacaa ggtactttat atacatctga gtaaacctca 3240
cacaattcta cagggaggta tttctatccc catttaacaa ataaggaaac gaagtccaag 3300
taaattaact tgcccaaggt cacacagata gtacctggca gaacaggaat ttaaacctaa 3360
atttgtccaa ctccaaaagc agccttctat ttgttataaa tgctgcctct cattatcaca 3420
tattttatta ttaacaacaa caaacatacc aattagctta agatacaata caaccagata 3480
atcatgatga caacagtaat tgttatacta ttataataaa atagatgttt tgtatgttac 3540
tataatcttg aatttgaata gaaatttgca tttctgaaag catgttcctg tcatctaata 3600
tgattctgta tctattaaaa tagtactaca tctagcccgg gctcgacatt gattattga 3659
<210> 7
<211> 2751
<212> ДНК
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<223> 1_68R2 (заполненный NcoI + XbaI)
<400> 7
gtacccccaa aagaaagaga tcctcaggta caaaatcccc ttccaagggt cttcaattca 60
atccaacaca tttgtaattg agtacctttt tcatgctaaa ctttcttgtt ttatacaaag 120
aaaatgaaga tgtggttatt gctcactagg agtttacaat cttgagaaaa gatgtaggcc 180
aagttacttg acctctaact acttcttttt ctcatccaca aagtgagaca attggaatat 240
tttaaaaaat atatatatat atttttatat atatgtgtat atatatattt tatatatatg 300
tgtatatata tattttatat atatgtgtat atatatattt tatatatatg tgtgtatata 360
tatactttat atatatgtgt gtatatatat tttatatata tgtgtatata tatatatttt 420
atatatatgt gtatatatat attttatata tatgtgtata tatatatttt atatatatgt 480
gtgtatatat atattttata tatatgtgtg tatatatatt ttatatatat gtgtatatat 540
atattttata tatatgtgta tatatatatt ttatatatgt gtgtatatat atattttata 600
tatgtgtgta tatatatatt ttatatatat gtgtgtatat atatatttta tatatatgtg 660
tgtatatata tattttatat atatgtgtgt atatatatat tttatatata tgtgtgtata 720
tatatatttt atatatatgt gtgtatatat atattttata tatatgtgtg tatatatata 780
ttttatatat atgtgtgtat atatatattt tatatatatg tgtgtatata tatattttat 840
atatatgtgt gtatatatat attttatata tatgtgtgta tatatatata ttttatatat 900
atgtgtgtat atatatatat tttatatata tatgtgtgta tatatatata ttttatatat 960
atatgtgtat atatatatat atatatattt tttttttgag acagagtctc gctctgtcgc 1020
ccaggctgga gtgcagtggc gcgatctcag ctcactgcaa cctccacctc ctgggttcaa 1080
gccgttctcc tgcctcagcc tcccgagtag ctgggactac aggcacgtgc caccacgtcc 1140
agctaatagt tttgtatctt tagtagagat ggggtttcac catattggtc aggatggtct 1200
cgatctcttg acctcatgat ccgcccacct cagcctccca aagtgctggg attacaggcg 1260
tgagccacca ctcccagcct aaaatatttt tatgatcttt ggccatattg ttcaatggac 1320
aaaatttagc cttcgaaggc aggccgattt gaggttaata ctacctttac cacttgatag 1380
ctatgtgacc ttggccatgt ggtttcaaca gtctgaacct cattttctct gtgtatgtgt 1440
ggtcctcctt acaagtttgt gaaaaatgtg aagtccttag ccatgatagc ccaatataac 1500
aggctaaatg ataataggtt tatgttcttt tcctttatat tctcagataa gcactgtcca 1560
agtttgaggt gttttgaggt ctcgcctgat ttggattgtt tgagtttatg ctattctttg 1620
aattctttga gctgttctga agcagtgtat catgaacaaa aacatcccca gttcagtcca 1680
aacccctggt tacatatcat tcttatgcca tgttataacc agtttgagag tgttccctct 1740
gttattgcat ttaagtttca gcctcacaca gaaattcagc agccaatttc taagccctaa 1800
gcataaaatc tggggtgggg gggggggatg gcctgaagag cagcattatg aatagcacca 1860
ttataattaa tgatctctca ggaagattta caatcacagg tagcagataa aacaaatagt 1920
actgcttctg cacttcccct ccttttattc gctatgaaat tttatgggaa atcagtccag 1980
tgaaaaatgt aagctcttaa tctttcccag aaatcctacc tcatttgatg aatactttga 2040
gggaatgaat tagagcattt ttttctttta tagtctactt cgcatttacg aagtgaggac 2100
ggtagcttag gctgcctggc caactgatga gaaggtcaga ggcattttta gagacctctg 2160
ttgtctttca ttcatgttca ttttccacaa ggcaagtaat ttccaacaaa tcagtgtctt 2220
cattagtaat aagattatta acaacaataa tagtcatagt aactattcag tgagagtcca 2280
ttatatatca ggcattctac aaggtacttt atatacatct gagtaaacct cacacaattc 2340
tacagggagg tatttctatc cccatttaac aaataaggaa acgaagtcca agtaaattaa 2400
cttgcccaag gtcacacaga tagtacctgg cagaacagga atttaaacct aaatttgtcc 2460
aactccaaaa gcagccttct atttgttata aatgctgcct ctcattatca catattttat 2520
tattaacaac aacaaacata ccaattagct taagatacaa tacaaccaga taatcatgat 2580
gacaacagta attgttatac tattataata aaatagatgt tttgtatgtt actataatct 2640
tgaatttgaa tagaaatttg catttctgaa agcatgttcc tgtcatctaa tatgattctg 2700
tatctattaa aatagtacta catctagccc gggctcgaca ttgattattg a 2751
<210> 8
<211> 2082
<212> ДНК
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<223> 1_6R2(заполненный HindIII)
<400> 8
tcgacatgga attacatggg catcttttgt ctgaaaccaa ttatgtatgt ttacgtatgt 60
gtatgtttat gcatatgtta taggtttaat atatattaat atatataata tataatatat 120
aaatattaat atgtattata taatatatat taatatatta tattatatta ctatataaat 180
aatattaata tattatatta aaatattaat aaatatatca tattaaatat tatattaatt 240
aaatattaat aaatatatta tattaatata tttatatatt aaacctataa catatgcata 300
tacttattta tatataacat gcatgtactt atttatatat acaatatata tttatatatt 360
atataatata ttatatgtat ttatatatta tatatcatat attatatgta tttatatatt 420
atatatcata taatatatat atttatatta tatatattat atgatatata atattatata 480
atgtattaat atatattaaa cctatattta taattctgga ctcactattt tgtttcattg 540
gtgtctgtgt gtatctaacc ctatgccaat aatgtactat cttaattacc atagctttat 600
agtaagctag cttgatatcg aattcctgca gcccggggga tccactagat ttggccatat 660
tgttcaatgg acaaaattta gccttcgaag gcaggccgat ttgaggttaa tactaccttt 720
accacttgat agctatgtga ccttggccat gtggtttcaa cagtctgaac ctcattttct 780
ctgtgtatgt gtggtcctcc ttacaagttt gtgaaaaatg tgaagtcctt agccatgata 840
gcccaatata acaggctaaa tgataatagg tttatgttct tttcctttat attctcagat 900
aagcactgtc caagtttgag gtgttttgag gtctcgcctg atttggattg tttgagttta 960
tgctattctt tgaattcttt gagctgttct gaagcagtgt atcatgaaca aaaacatccc 1020
cagttcagtc caaacccctg gttacatatc attcttatgc catgttataa ccagtttgag 1080
agtgttccct ctgttattgc atttaagttt cagcctcaca cagaaattca gcagccaatt 1140
tctaagccct aagcataaaa tctggggtgg ggggggggga tggcctgaag agcagcatta 1200
tgaatagcac cattataatt aatgatctct caggaagatt tacaatcaca ggtagcagat 1260
aaaacaaata gtactgcttc tgcacttccc ctccttttat tcgctatgaa attttatggg 1320
aaatcagtcc agtgaaaaat gtaagctctt aatctttccc agaaatccta cctcatttga 1380
tgaatacttt gagggaatga attagagcat ttttttcttt tatagtctac ttcgcattta 1440
cgaagtgagg acggtagctt aggctgcctg gccaactgat gagaaggtca gaggcatttt 1500
tagagacctc tgttgtcttt cattcatgtt cattttccac aaggcaagta atttccaaca 1560
aatcagtgtc ttcattagta ataagattat taacaacaat aatagtcata gtaactattc 1620
agtgagagtc cattatatat caggcattct acaaggtact ttatatacat ctgagtaaac 1680
ctcacacaat tctacaggga ggtatttcta tccccattta acaaataagg aaacgaagtc 1740
caagtaaatt aacttgccca aggtcacaca gatagtacct ggcagaacag gaatttaaac 1800
ctaaatttgt ccaactccaa aagcagcctt ctatttgtta taaatgctgc ctctcattat 1860
cacatatttt attattaaca acaacaaaca taccaattag cttaagatac aatacaacca 1920
gataatcatg atgacaacag taattgttat actattataa taaaatagat gttttgtatg 1980
ttactataat cttgaatttg aatagaaatt tgcatttctg aaagcatgtt cctgtcatct 2040
aatatgattc tgtatctatt aaaatagtac tacatctagc cc 2082
<210> 9
<211> 3345
<212> ДНК
<213> Homo sapiens
<220>
<221> X_S29(заполненный 2*HindIII, SalI)
<222> (1)..(3345)
<220>
<221> misc_feature
<222> (1)..(3345)
<400> 9
gatcccttta taaaaccaca atataatgga gtgctataat ttcaaacagt gtttggtctg 60
ctggcagagt ggtcattcta acagcagtca cagtagagta gaaataagac tgcagtatat 120
ctaaggcaaa aagctgaggt ttcaggagct tgaaggtaaa gaggaagaaa gaaatgggaa 180
tgggaattgg aaagacaaat atcgttaaga gaaaattgct tttaggagag gggaaagaat 240
ctatgtgtac ttaagactat ggaatcaatc ccatttaagc tgggaaacta gtttcatata 300
taactaataa attttattta cagaatatct atttacctga tctaggcttc aagccaaagg 360
gactgtgtga aaaaccatca gttctgtcat attcctaaaa aaaaattaaa aagttaaaaa 420
taaataaata ataaaacttc ttttctttca aaataatcaa ggtgcttatt cacatccatt 480
ccaatttggg gaaatactta ttttcctatg attagcgaag agaaaagtaa cttgcatttc 540
aattcaagtt gatacatgtc acttttaaga ggtcaactaa tatttgctag ttgagctaac 600
catataggct ttaaatactt tcatagtaga aagaaaatga aaatcattag tgaactgtat 660
aaaatagatc atactttttg aaagaatcag actgaagttt ccgaaaaaaa gaagtaagct 720
agcttcaatg aaaaggtaag tgaatttagc atttactcag catctactat ggacttaaca 780
cctaacagta gataatctga aggcaaacat atttgtatag ggactgcaga atgatagatg 840
ataaatatca tctcttctat ttgaatgaat attttttcaa atctttcaca cacagtggtt 900
tgctatggaa agatttgtag tacattaaac aaatctgaag atggagttag aaagctagct 960
taggctatgt tttgagcaca acatataatt tctctgtgat tgtttcttca tctttcaaat 1020
gaggttactg tgaagattaa atgagataac taaatgatga taaaataatg taatcttagc 1080
agcaccttat ttaatctgtg caacaactct gtgaagtgag tagggctcag cttcagtcac 1140
ttctctgcca tttattaact aagatagttt ggaaagttac ccatctcttc agctgtaaaa 1200
tgatgaggat catacctatt ttatggggct gcttttaggt acaaatatac aggcaagcac 1260
tttgttaata ctaaagcatt acaccaatta gttttactct tttccattca cacatgaaat 1320
taatgtaatc agaattctgt agattaccta aatcttctgt taacacgtga tatgcagttc 1380
aggttaaatg tcagttgagt taccaaagca catacatact caccacccta tccaaatcta 1440
caagcctccc agtttgtctt cactattttg gttaaattaa tatgaattcc tagatgaaaa 1500
tttcactgat ccaaatgaaa taaaaaatat attacaaaac tcacacctgt aatctcaaca 1560
ttttgggagg ccaaggcagg tagatcactt gaggccagga gttcaagacc agcctgatca 1620
acatggtgaa accctgtctc tactaaaaat acaaaaatta gccaggtgtg gtggcatgtg 1680
cctgtagtcc tacctactcg ggaggctgag gcacaagaat cgcttgaatg tgggaggtgg 1740
aggttgcagt gacctgagat cgtgccactg cactccagcc taggcaacag agtgagatca 1800
tgtgtcatat atatatatat atatatatat atatatatat atatatacac acacacacac 1860
atatatatat acacatatat atacgtatat atatatatgt atatatatac atatatatac 1920
atatatatat atacgtatat atatacgtat atatatatca atgtaaatta tttgggaaat 1980
ttggtatgaa tagtcttccc tgtgaacaca gatcataaaa tcatatatca agcagacaaa 2040
taagtagtag tcacttatat gcttatactt gtaacttaaa gtaaaagaat tacaaaagca 2100
tatgacaaag actaatttta agatatccta atttaaattg ttttctaaaa gtgtgtatac 2160
cattttacct atcatatgaa taatttagaa acatgtttat aaaattaatg tccaaatcca 2220
ttcaaaagtt ttgtaatgca gatcacccac aacaacaaag aatcctagcc tattaaaaaa 2280
gcaacaccac ctacatataa tgaaatatta gcagcatcta tgtaaccaaa gttacacagt 2340
gaatttgggc catccaacac tttgagcaaa gtgttgaatt catcaaatga atgtgtaatc 2400
atttacttac taatgccaat acactttaag gtaatcttaa gtagaagaga tagagtttag 2460
aattttttaa atttatctct tgttgtaaag caatagactt gaataaataa attagaagaa 2520
tcagtcattc aagccaccag agtatttgat cgagatttca caaactctaa ctttctgata 2580
cccattctcc caaaaacgtg taacctcctg tcgataggaa caacccactg cagggatgtt 2640
tctcgtggaa aaaggaaatt tcttttgcat tggtttcaga cctaactggt tacaagaaaa 2700
accaaaggcc attgcacaat gctgaagtac ttttttcaaa tttaaaattt gaaagttgtt 2760
cttaaaatct atcatttatt ttaaaatacg gatgaatgag aaagcataga tttgataaag 2820
tgaattcttt tctgcaatct acagacactt ccaaaaatca ctacagacac tacagacact 2880
acagaaaatc ataaataaac aagtgctagt atcaatattt ttaccaaaaa atggcattct 2940
tagaattttt tataggctag aaggtttgta caaactaatc tgccacggat tttaaaatat 3000
gagtgaataa attatattgc aaaaaaaatc aggttacaga gaactggcaa ggaagactct 3060
tatgtaaaac acagaaaaca tacaaaacgt atttttaaga caaataaaaa cagaacttgt 3120
acctcagatg atactggaga ttgtgttgac atattagcat tatcactgtc ttgctaaaac 3180
ataaaaataa aaagatggaa gatgaaatta caatacaaat gatgatttaa acatataaaa 3240
ggaaaataaa aattgttctg accaactact aaaggaagac ctactaaaga tatgccatcc 3300
agcacattgc cactctacat gtggtctgta aaccagcagc atagg 3345
<210> 10
<211> 892
<212> ДНК
<213> Homo sapiens
<220>
<221> misc_feature
<222> (1)..(892)
<223> X_S29R3
<400> 10
aattcaacac tttgctcaaa gtgttggatg gcccaaattc actgtgtaac tttggttaca 60
tagatgctgc taatatttca ttatatgtag gtggtgttgc ttttttaata ggctaggatt 120
ctttgttgtt gtgggtgatc tgcattacaa aacttttgaa tggatttgga cattaatttt 180
ataaacatgt ttctaaatta ttcatatgat aggtaaaatg gtatacacac ttttagaaaa 240
caatttaaat taggatatct taaaattagt ctttgtcata tgcttttgta attcttttac 300
tttaagttac aagtataagc atataagtga ctactactta tttgtctgct tgatatatga 360
ttttatgatc tgtgttcaca gggaagacta ttcataccaa atttcccaaa taatttacat 420
tgatatatat atacgtatat atatacgtat atatatatat gtatatatat gtatatatat 480
acatatatat atatacgtat atatatgtgt atatatatat gtgtgtgtgt gtgtatatat 540
atatatatat atatatatat atatatatat atatgacaca tgatctcact ctgttgccta 600
ggctggagtg cagtggcacg atctcaggtc actgcaacct ccacctccca cattcaagcg 660
attcttgtgc ctcagcctcc cgagtaggta ggactacagg cacatgccac cacacctggc 720
taatttttgt atttttagta gagacagggt ttcaccatgt tgatcaggct ggtcttgaac 780
tcctggcctc aagtgatcta cctgccttgg cctcccaaaa tgttgagatt acaggtgtga 840
gttttgtaat atatttttta tttcatttgg atcagtgaaa ttttcatcta gg 892
<210> 11
<211> 1173
<212> ДНК
<213> Homo sapiens
<220>
<221> Промотор C_GAPDH
<222> (1)..(363)
<223> Энхансер немедленно-ранних генов CMV человека
<400> 11
ctagttatta atagtaatca attacggggt cattagttca tagcccatat atggagttcc 60
gcgttacata acttacggta aatggcccgc ctggctgacc gcccaacgac ccccgcccat 120
tgacgtcaat aatgacgtat gttcccatag taacgccaat agggactttc cattgacgtc 180
aatgggtgga ctatttacgg taaactgccc acttggcagt acatcaagtg tatcatatgc 240
caagtacgcc ccctattgac gtcaatgacg gtaaatggcc cgcctggcat tatgcccagt 300
acatgacctt atgggacttt cctacttggc agtacatcta cgtattagtc atcgctatta 360
ccatgacctc gaggagaagt tccccaactt tcccgcctct cagcctttga aagaaagaaa 420
ggggaggggg caggccgcgt gcagccgcga gcggtgctgg gctccggctc caattcccca 480
tctcagtcgt tcccaaagtc ctcctgtttc atccaagcgt gtaagggtcc ccgtccttga 540
ctccctagtg tcctgctgcc cacagtccag tcctgggaac cagcaccgat cacctcccat 600
cgggccaatc tcagtccctt cccccctacg tcggggccca cacgctcggt gcgtgcccag 660
ttgaaccagg cggctgcgga aaaaaaaaag cggggagaaa gtagggcccg gctactagcg 720
gttttacggg cgcacgtagc tcaggcctca agaccttggg ctgggactgg ctgagcctgg 780
cgggaggcgg ggtccgagtc accgcctgcc gccgcgcccc cggtttctat aaattgagcc 840
cgcagcctcc cgcttcgctc tctgctcctc ctgttcgaca gtcagccgca tcttcttttg 900
cgtcgccagg tgaagacggg cggagagaaa cccgggaggc tagggacggc ctgaaggcgg 960
caggggcggg cgcaggccgg atgtgttcgc gccgctgcgg ggtgggcccg ggcggcctcc 1020
gcattgcagg ggcgggcgga ggacgtgatg cggcgcgggc tgggcatgga ggcctggtgg 1080
gggaggggag gggaggcgtg tgtgtcggcc ggggccacta ggcgctcact gttctctccc 1140
tccgcgcagc cgagccacat cgctcagaca cca 1173
<210> 12
<211> 411
<212> ДНК
<213> Homo sapiens
<220>
<221> misc_feature
<223> hSRP14
<400> 12
atggtgttgt tggagagcga gcagttcctg acggagctga ccagactttt ccagaagtgc 60
cggacgtcgg gcagcgtcta tatcaccttg aagaagtatg acggtcgaac caaacccatt 120
ccaaagaagg gtactgtgga gggctttgag cccgcagaca acaagtgtct gttaagagct 180
accgatggga agaagaagat cagcactgtg gtgagctcca aggaagtgaa taagtttcag 240
atggcttatt caaacctcct tagagctaac atggatgggc tgaagaagag agacaaaaag 300
aacaaaacta agaagaccaa agcagcagca gcagcagcag cagcagcacc tgccgcagca 360
gcaacagcac caacaacagc agcaacaaca gcagcaacag cagcacagta a 411
<210> 13
<211> 136
<212> ПРТ
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<223> hSRP14
<400> 13
Met Val Leu Leu Glu Ser Glu Gln Phe Leu Thr Glu Leu Thr Arg Leu
1 5 10 15
Phe Gln Lys Cys Arg Thr Ser Gly Ser Val Tyr Ile Thr Leu Lys Lys
20 25 30
Tyr Asp Gly Arg Thr Lys Pro Ile Pro Lys Lys Gly Thr Val Glu Gly
35 40 45
Phe Glu Pro Ala Asp Asn Lys Cys Leu Leu Arg Ala Thr Asp Gly Lys
50 55 60
Lys Lys Ile Ser Thr Val Val Ser Ser Lys Glu Val Asn Lys Phe Gln
65 70 75 80
Met Ala Tyr Ser Asn Leu Leu Arg Ala Asn Met Asp Gly Leu Lys Lys
85 90 95
Arg Asp Lys Lys Asn Lys Thr Lys Lys Thr Lys Ala Ala Ala Ala Ala
100 105 110
Ala Ala Ala Ala Pro Ala Ala Ala Ala Thr Ala Pro Thr Thr Ala Ala
115 120 125
Thr Thr Ala Ala Thr Ala Ala Gln
130 135
<210> 14
<211> 1431
<212> ДНК
<213> Homo sapiens
<220>
<221> misc_feature
<223> hSEC61A1
<400> 14
atggcaatca aatttctgga agtcatcaag cccttctgtg tcatcctgcc ggaaattcag 60
aagccagaga ggaagattca gtttaaggag aaagtgctgt ggaccgctat caccctcttt 120
atcttcttag tgtgctgcca gattcccctg tttgggatca tgtcttcaga ttcagctgac 180
cctttctatt ggatgagagt gattctagcc tctaacagag gcacattgat ggagctaggg 240
atctctccta ttgtcacgtc tggccttata atgcaactct tggctggcgc caagataatt 300
gaagttggtg acaccccaaa agaccgagct ctcttcaacg gagcccaaaa gttatttggc 360
atgatcatta ctatcggcca gtctatcgtg tatgtgatga ccgggatgta tggggaccct 420
tctgaaatgg gtgctggaat ttgcctgcta atcaccattc agctctttgt tgctggctta 480
attgtcctac ttttggatga actcctgcaa aaaggatatg gccttggctc tggtatttct 540
ctcttcattg caactaacat ctgtgaaacc atcgtatgga aggcattcag ccccactact 600
gtcaacactg gccgaggaat ggaatttgaa ggtgctatca tcgcactttt ccatctgctg 660
gccacacgca cagacaaggt ccgagccctt cgggaggcgt tctaccgcca gaatcttccc 720
aacctcatga atctcatcgc caccatcttt gtctttgcag tggtcatcta tttccagggc 780
ttccgagtgg acctgccaat caagtcggcc cgctaccgtg gccagtacaa cacctatccc 840
atcaagctct tctatacgtc caacatcccc atcatcctgc agtctgccct ggtgtccaac 900
ctttatgtca tctcccaaat gctctcagct cgcttcagtg gcaacttgct ggtcagcctg 960
ctgggcacct ggtcggacac gtcttctggg ggcccagcac gtgcttatcc agttggtggc 1020
ctttgctatt acctgtcccc tccagaatct tttggctccg tgttagaaga cccggtccat 1080
gcagttgtat acatagtgtt catgctgggc tcctgtgcat tcttctccaa aacgtggatt 1140
gaggtctcag gttcctctgc caaagatgtt gcaaagcagc tgaaggagca gcagatggtg 1200
atgagaggcc accgagagac ctccatggtc catgaactca accggtacat ccccacagcc 1260
gcggcctttg gtgggctgtg catcggggcc ctctcggtcc tggctgactt cctaggcgcc 1320
attgggtctg gaaccgggat cctgctcgca gtcacaatca tctaccagta ctttgagatc 1380
ttcgttaagg agcaaagcga ggttggcagc atgggggccc tgctcttctg a 1431
<210> 15
<211> 476
<212> ПРТ
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<223> hSEC61A1
<400> 15
Met Ala Ile Lys Phe Leu Glu Val Ile Lys Pro Phe Cys Val Ile Leu
1 5 10 15
Pro Glu Ile Gln Lys Pro Glu Arg Lys Ile Gln Phe Lys Glu Lys Val
20 25 30
Leu Trp Thr Ala Ile Thr Leu Phe Ile Phe Leu Val Cys Cys Gln Ile
35 40 45
Pro Leu Phe Gly Ile Met Ser Ser Asp Ser Ala Asp Pro Phe Tyr Trp
50 55 60
Met Arg Val Ile Leu Ala Ser Asn Arg Gly Thr Leu Met Glu Leu Gly
65 70 75 80
Ile Ser Pro Ile Val Thr Ser Gly Leu Ile Met Gln Leu Leu Ala Gly
85 90 95
Ala Lys Ile Ile Glu Val Gly Asp Thr Pro Lys Asp Arg Ala Leu Phe
100 105 110
Asn Gly Ala Gln Lys Leu Phe Gly Met Ile Ile Thr Ile Gly Gln Ser
115 120 125
Ile Val Tyr Val Met Thr Gly Met Tyr Gly Asp Pro Ser Glu Met Gly
130 135 140
Ala Gly Ile Cys Leu Leu Ile Thr Ile Gln Leu Phe Val Ala Gly Leu
145 150 155 160
Ile Val Leu Leu Leu Asp Glu Leu Leu Gln Lys Gly Tyr Gly Leu Gly
165 170 175
Ser Gly Ile Ser Leu Phe Ile Ala Thr Asn Ile Cys Glu Thr Ile Val
180 185 190
Trp Lys Ala Phe Ser Pro Thr Thr Val Asn Thr Gly Arg Gly Met Glu
195 200 205
Phe Glu Gly Ala Ile Ile Ala Leu Phe His Leu Leu Ala Thr Arg Thr
210 215 220
Asp Lys Val Arg Ala Leu Arg Glu Ala Phe Tyr Arg Gln Asn Leu Pro
225 230 235 240
Asn Leu Met Asn Leu Ile Ala Thr Ile Phe Val Phe Ala Val Val Ile
245 250 255
Tyr Phe Gln Gly Phe Arg Val Asp Leu Pro Ile Lys Ser Ala Arg Tyr
260 265 270
Arg Gly Gln Tyr Asn Thr Tyr Pro Ile Lys Leu Phe Tyr Thr Ser Asn
275 280 285
Ile Pro Ile Ile Leu Gln Ser Ala Leu Val Ser Asn Leu Tyr Val Ile
290 295 300
Ser Gln Met Leu Ser Ala Arg Phe Ser Gly Asn Leu Leu Val Ser Leu
305 310 315 320
Leu Gly Thr Trp Ser Asp Thr Ser Ser Gly Gly Pro Ala Arg Ala Tyr
325 330 335
Pro Val Gly Gly Leu Cys Tyr Tyr Leu Ser Pro Pro Glu Ser Phe Gly
340 345 350
Ser Val Leu Glu Asp Pro Val His Ala Val Val Tyr Ile Val Phe Met
355 360 365
Leu Gly Ser Cys Ala Phe Phe Ser Lys Thr Trp Ile Glu Val Ser Gly
370 375 380
Ser Ser Ala Lys Asp Val Ala Lys Gln Leu Lys Glu Gln Gln Met Val
385 390 395 400
Met Arg Gly His Arg Glu Thr Ser Met Val His Glu Leu Asn Arg Tyr
405 410 415
Ile Pro Thr Ala Ala Ala Phe Gly Gly Leu Cys Ile Gly Ala Leu Ser
420 425 430
Val Leu Ala Asp Phe Leu Gly Ala Ile Gly Ser Gly Thr Gly Ile Leu
435 440 445
Leu Ala Val Thr Ile Ile Tyr Gln Tyr Phe Glu Ile Phe Val Lys Glu
450 455 460
Gln Ser Glu Val Gly Ser Met Gly Ala Leu Leu Phe
465 470 475
<210> 16
<211> 291
<212> ДНК
<213> Homo sapiens
<220>
<221> misc_feature
<223> hSEC61B
<400> 16
atgcctggtc cgacccccag tggcactaac gtgggatcct cagggcgctc tcccagcaaa 60
gcagtggccg cccgggcggc gggatccact gtccggcaga ggaaaaatgc cagctgtggg 120
acaaggagtg caggccgcac aacctcggca ggcaccgggg ggatgtggcg attctacaca 180
gaagattcac ctgggctcaa agttggccct gttccagtat tggttatgag tcttctgttc 240
atcgcttctg tatttatgtt gcacatttgg ggcaagtaca ctcgttcgta g 291
<210> 17
<211> 96
<212> ПРТ
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<223> hSEC61B
<400> 17
Met Pro Gly Pro Thr Pro Ser Gly Thr Asn Val Gly Ser Ser Gly Arg
1 5 10 15
Ser Pro Ser Lys Ala Val Ala Ala Arg Ala Ala Gly Ser Thr Val Arg
20 25 30
Gln Arg Lys Asn Ala Ser Cys Gly Thr Arg Ser Ala Gly Arg Thr Thr
35 40 45
Ser Ala Gly Thr Gly Gly Met Trp Arg Phe Tyr Thr Glu Asp Ser Pro
50 55 60
Gly Leu Lys Val Gly Pro Val Pro Val Leu Val Met Ser Leu Leu Phe
65 70 75 80
Ile Ala Ser Val Phe Met Leu His Ile Trp Gly Lys Tyr Thr Arg Ser
85 90 95
<210> 18
<211> 207
<212> ДНК
<213> Homo sapiens
<220>
<221> misc_feature
<223> hSEC61G
<400> 18
atggatcagg taatgcagtt tgttgagcca agtcggcagt ttgtaaagga ctccattcgg 60
ctggttaaaa gatgcactaa acctgataga aaagaattcc agaagattgc catggcaaca 120
gcaataggat ttgctataat gggattcatt ggcttctttg tgaaattgat ccatattcct 180
attaataaca tcattgttgg tggctga 207
<210> 19
<211> 68
<212> ПРТ
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<223> hSEC61G
<400> 19
Met Asp Gln Val Met Gln Phe Val Glu Pro Ser Arg Gln Phe Val Lys
1 5 10 15
Asp Ser Ile Arg Leu Val Lys Arg Cys Thr Lys Pro Asp Arg Lys Glu
20 25 30
Phe Gln Lys Ile Ala Met Ala Thr Ala Ile Gly Phe Ala Ile Met Gly
35 40 45
Phe Ile Gly Phe Phe Val Lys Leu Ile His Ile Pro Ile Asn Asn Ile
50 55 60
Ile Val Gly Gly
65
<210> 20
<211> 1515
<212> ДНК
<213> Homo sapiens
<220>
<221> misc_feature
<223> hSRP54
<400> 20
atggttctag cagaccttgg aagaaaaata acatcagcat tacgctcgtt gagcaatgcc 60
accattatca atgaagaggt attgaatgct atgctaaaag aagtctgtac cgctttgttg 120
gaagcagatg ttaatattaa actagtgaag caactaagag aaaatgttaa gtctgctatt 180
gatcttgaag agatggcatc tggtcttaac aaaagaaaaa tgattcagca tgctgtattt 240
aaagaacttg tgaagcttgt agaccctgga gttaaggcat ggacacccac taaaggaaaa 300
caaaatgtga ttatgtttgt tggattgcaa gggagtggta aaacaacaac atgttcaaag 360
ctagcatatt attaccagag gaaaggttgg aagacctgtt taatatgtgc agacacattc 420
agagcagggg cttttgacca actaaaacag aatgctacca aagcaagaat tccattttat 480
ggaagctata cagaaatgga tcctgtcatc attgcttctg aaggagtaga gaaatttaaa 540
aatgaaaatt ttgaaattat tattgttgat acaagtggcc gccacaaaca agaagactct 600
ttgtttgaag aaatgcttca agttgctaat gctatacaac ctgataacat tgtttatgtg 660
atggatgcct ccattgggca ggcttgtgaa gcccaggcta aggcttttaa agataaagta 720
gatgtagcct cagtaatagt gacaaaactt gatggccatg caaaaggagg tggtgcactc 780
agtgcagtcg ctgccacaaa aagtccgatt attttcattg gtacagggga acatatagat 840
gactttgaac ctttcaaaac acagcctttt attagcaaac ttcttggtat gggcgacatt 900
gaaggactga tagataaagt caacgagttg aagttggatg acaatgaagc acttatagag 960
aagttgaaac atggtcagtt tacgttgcga gacatgtatg agcaatttca aaatatcatg 1020
aaaatgggcc ccttcagtca gatcttgggg atgatccctg gttttgggac agattttatg 1080
agcaaaggaa atgaacagga gtcaatggca aggctaaaga aattaatgac aataatggat 1140
agtatgaatg atcaagaact agacagtacg gatggtgcca aagtttttag taaacaacca 1200
ggaagaatcc aaagagtagc aagaggatcg ggtgtatcaa caagagatgt tcaagaactt 1260
ttgacacaat ataccaagtt tgcacagatg gtaaaaaaga tgggaggtat caaaggactt 1320
ttcaaaggtg gcgacatgtc taagaatgtg agccagtcac agatggcaaa attgaaccaa 1380
caaatggcca aaatgatgga tcctagggtt cttcatcaca tgggtggtat ggcaggactt 1440
cagtcaatga tgaggcagtt tcaacagggt gctgctggca acatgaaagg catgatggga 1500
ttcaataata tgtaa 1515
<210> 21
<211> 504
<212> ПРТ
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<223> hSRP54
<400> 21
Met Val Leu Ala Asp Leu Gly Arg Lys Ile Thr Ser Ala Leu Arg Ser
1 5 10 15
Leu Ser Asn Ala Thr Ile Ile Asn Glu Glu Val Leu Asn Ala Met Leu
20 25 30
Lys Glu Val Cys Thr Ala Leu Leu Glu Ala Asp Val Asn Ile Lys Leu
35 40 45
Val Lys Gln Leu Arg Glu Asn Val Lys Ser Ala Ile Asp Leu Glu Glu
50 55 60
Met Ala Ser Gly Leu Asn Lys Arg Lys Met Ile Gln His Ala Val Phe
65 70 75 80
Lys Glu Leu Val Lys Leu Val Asp Pro Gly Val Lys Ala Trp Thr Pro
85 90 95
Thr Lys Gly Lys Gln Asn Val Ile Met Phe Val Gly Leu Gln Gly Ser
100 105 110
Gly Lys Thr Thr Thr Cys Ser Lys Leu Ala Tyr Tyr Tyr Gln Arg Lys
115 120 125
Gly Trp Lys Thr Cys Leu Ile Cys Ala Asp Thr Phe Arg Ala Gly Ala
130 135 140
Phe Asp Gln Leu Lys Gln Asn Ala Thr Lys Ala Arg Ile Pro Phe Tyr
145 150 155 160
Gly Ser Tyr Thr Glu Met Asp Pro Val Ile Ile Ala Ser Glu Gly Val
165 170 175
Glu Lys Phe Lys Asn Glu Asn Phe Glu Ile Ile Ile Val Asp Thr Ser
180 185 190
Gly Arg His Lys Gln Glu Asp Ser Leu Phe Glu Glu Met Leu Gln Val
195 200 205
Ala Asn Ala Ile Gln Pro Asp Asn Ile Val Tyr Val Met Asp Ala Ser
210 215 220
Ile Gly Gln Ala Cys Glu Ala Gln Ala Lys Ala Phe Lys Asp Lys Val
225 230 235 240
Asp Val Ala Ser Val Ile Val Thr Lys Leu Asp Gly His Ala Lys Gly
245 250 255
Gly Gly Ala Leu Ser Ala Val Ala Ala Thr Lys Ser Pro Ile Ile Phe
260 265 270
Ile Gly Thr Gly Glu His Ile Asp Asp Phe Glu Pro Phe Lys Thr Gln
275 280 285
Pro Phe Ile Ser Lys Leu Leu Gly Met Gly Asp Ile Glu Gly Leu Ile
290 295 300
Asp Lys Val Asn Glu Leu Lys Leu Asp Asp Asn Glu Ala Leu Ile Glu
305 310 315 320
Lys Leu Lys His Gly Gln Phe Thr Leu Arg Asp Met Tyr Glu Gln Phe
325 330 335
Gln Asn Ile Met Lys Met Gly Pro Phe Ser Gln Ile Leu Gly Met Ile
340 345 350
Pro Gly Phe Gly Thr Asp Phe Met Ser Lys Gly Asn Glu Gln Glu Ser
355 360 365
Met Ala Arg Leu Lys Lys Leu Met Thr Ile Met Asp Ser Met Asn Asp
370 375 380
Gln Glu Leu Asp Ser Thr Asp Gly Ala Lys Val Phe Ser Lys Gln Pro
385 390 395 400
Gly Arg Ile Gln Arg Val Ala Arg Gly Ser Gly Val Ser Thr Arg Asp
405 410 415
Val Gln Glu Leu Leu Thr Gln Tyr Thr Lys Phe Ala Gln Met Val Lys
420 425 430
Lys Met Gly Gly Ile Lys Gly Leu Phe Lys Gly Gly Asp Met Ser Lys
435 440 445
Asn Val Ser Gln Ser Gln Met Ala Lys Leu Asn Gln Gln Met Ala Lys
450 455 460
Met Met Asp Pro Arg Val Leu His His Met Gly Gly Met Ala Gly Leu
465 470 475 480
Gln Ser Met Met Arg Gln Phe Gln Gln Gly Ala Ala Gly Asn Met Lys
485 490 495
Gly Met Met Gly Phe Asn Asn Met
500
<210> 22
<211> 249
<212> ДНК
<213> Homo sapiens
<220>
<221> misc_feature
<223> hSRP9
<400> 22
atgccgcagt accagacctg ggaggagttc agccgcgctg ccgagaagct ttacctcgct 60
gaccctatga aggcacgtgt ggttctcaaa tataggcatt ctgatgggaa cttgtgtgtt 120
aaagtaacag atgatttagt tagacagtgt cttgctctat tgctcaggct gcagtgcagt 180
ggcatgatca tagctcactg catcctcgac ctcctgggct caagcggtcc tcttgcttca 240
gcctcctga 249
<210> 23
<211> 82
<212> ПРТ
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<223> hSRP9
<400> 23
Met Pro Gln Tyr Gln Thr Trp Glu Glu Phe Ser Arg Ala Ala Glu Lys
1 5 10 15
Leu Tyr Leu Ala Asp Pro Met Lys Ala Arg Val Val Leu Lys Tyr Arg
20 25 30
His Ser Asp Gly Asn Leu Cys Val Lys Val Thr Asp Asp Leu Val Arg
35 40 45
Gln Cys Leu Ala Leu Leu Leu Arg Leu Gln Cys Ser Gly Met Ile Ile
50 55 60
Ala His Cys Ile Leu Asp Leu Leu Gly Ser Ser Gly Pro Leu Ala Ser
65 70 75 80
Ala Ser
<210> 24
<211> 1917
<212> ДНК
<213> Homo sapiens
<220>
<221> misc_feature
<223> hSRPR-альфа
<400> 24
atgctcgact tcttcaccat tttctccaag ggcgggcttg tgctctggtg cttccagggc 60
gttagcgact catgcaccgg acccgttaac gcgttgattc gttccgtgct gctgcaggaa 120
cggggaggta acaactcctt cacccatgag gcactcacac tcaagtataa actggacaac 180
cagtttgagc tggtgtttgt ggttggtttt cagaagatcc tgacactgac atatgtagac 240
aaattgatag atgacgtgca tcggctgttt cgggacaagt accgcacaga gatccaacag 300
caaagtgctt taagtttatt aaatggcact tttgatttcc aaaatgactt cctgcggctc 360
cttcgtgaag cagaggagag cagtaagatc cgtgctccca ctaccatgaa gaaatttgaa 420
gattctgaaa aggccaagaa acctgtgagg tccatgattg agacacgggg ggaaaagccc 480
aaggaaaaag caaagaatag caaaaaaaag ggggccaaga aggaaggttc tgatggtcct 540
ttggctacca gcaaaccagt ccctgcagaa aagtcaggtc ttccagtggg tcctgagaac 600
ggagtagaac tttccaaaga ggagctgatc cgcaggaagc gcgaggagtt cattcagaag 660
catgggaggg gtatggagaa gtccaacaag tccacgaagt cagatgctcc aaaggagaag 720
ggcaaaaaag caccccgggt gtgggaactg ggtggctgtg ctaacaaaga agtgttggat 780
tacagtactc ccaccaccaa tggaacccct gaggctgcct tgtctgagga catcaacctg 840
attcgaggga ctgggtctgg ggggcagctt caggatctgg actgcagcag ctctgatgac 900
gaaggggctg ctcaaaactc taccaaacct agtgcgacca agggaacact gggtggcatg 960
tttggtatgc tgaagggcct tgtgggttca aagagcttga gtcgtgaaga catggaatct 1020
gtgctggaca agatgcgtga tcatctcatt gctaagaacg tggctgcaga cattgccgtc 1080
cagctctgtg aatctgttgc caacaagttg gaagggaagg tgatggggac gttcagcacg 1140
gtgacttcca cagtaaagca agccctacag gagtccctgg tgcagattct gcagccacag 1200
cgtcgtgtag acatgctccg ggacatcatg gatgcccagc gtcgccagcg cccttatgtc 1260
gtcaccttct gcggcgttaa tggagtgggg aaatctacta atcttgccaa gatttccttc 1320
tggttgttag agaatggctt cagtgtcctc attgctgcct gtgatacatt tcgtgctggg 1380
gccgtggagc agctgcgtac acacacccgg cgtttgagtg ccctacaccc tccagagaag 1440
catggtggcc gcaccatggt gcagttgttt gaaaagggct atggcaagga tgctgctggc 1500
attgccatgg aagccattgc ttttgcacgt aaccaaggct ttgacgtggt gctggtggac 1560
acggcaggcc gcatgcaaga caatgcccct ctgatgactg ccctggccaa actcattact 1620
gtcaatacac ctgatttggt gctgtttgta ggagaagcct tagtaggcaa tgaagccgtg 1680
gaccagctgg tcaagttcaa cagagccttg gctgaccatt ctatggctca gacacctcgg 1740
ctcattgatg gcattgttct taccaaattt gataccattg atgacaaggt gggagctgct 1800
atttctatga cgtacatcac aagcaaaccc atcgtctttg tgggcaccgg ccagacctac 1860
tgtgacctac gcagcctcaa tgccaaggct gtggtggctg ccctcatgaa ggcttaa 1917
<210> 25
<211> 638
<212> ПРТ
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<223> hSRPR-альфа
<400> 25
Met Leu Asp Phe Phe Thr Ile Phe Ser Lys Gly Gly Leu Val Leu Trp
1 5 10 15
Cys Phe Gln Gly Val Ser Asp Ser Cys Thr Gly Pro Val Asn Ala Leu
20 25 30
Ile Arg Ser Val Leu Leu Gln Glu Arg Gly Gly Asn Asn Ser Phe Thr
35 40 45
His Glu Ala Leu Thr Leu Lys Tyr Lys Leu Asp Asn Gln Phe Glu Leu
50 55 60
Val Phe Val Val Gly Phe Gln Lys Ile Leu Thr Leu Thr Tyr Val Asp
65 70 75 80
Lys Leu Ile Asp Asp Val His Arg Leu Phe Arg Asp Lys Tyr Arg Thr
85 90 95
Glu Ile Gln Gln Gln Ser Ala Leu Ser Leu Leu Asn Gly Thr Phe Asp
100 105 110
Phe Gln Asn Asp Phe Leu Arg Leu Leu Arg Glu Ala Glu Glu Ser Ser
115 120 125
Lys Ile Arg Ala Pro Thr Thr Met Lys Lys Phe Glu Asp Ser Glu Lys
130 135 140
Ala Lys Lys Pro Val Arg Ser Met Ile Glu Thr Arg Gly Glu Lys Pro
145 150 155 160
Lys Glu Lys Ala Lys Asn Ser Lys Lys Lys Gly Ala Lys Lys Glu Gly
165 170 175
Ser Asp Gly Pro Leu Ala Thr Ser Lys Pro Val Pro Ala Glu Lys Ser
180 185 190
Gly Leu Pro Val Gly Pro Glu Asn Gly Val Glu Leu Ser Lys Glu Glu
195 200 205
Leu Ile Arg Arg Lys Arg Glu Glu Phe Ile Gln Lys His Gly Arg Gly
210 215 220
Met Glu Lys Ser Asn Lys Ser Thr Lys Ser Asp Ala Pro Lys Glu Lys
225 230 235 240
Gly Lys Lys Ala Pro Arg Val Trp Glu Leu Gly Gly Cys Ala Asn Lys
245 250 255
Glu Val Leu Asp Tyr Ser Thr Pro Thr Thr Asn Gly Thr Pro Glu Ala
260 265 270
Ala Leu Ser Glu Asp Ile Asn Leu Ile Arg Gly Thr Gly Ser Gly Gly
275 280 285
Gln Leu Gln Asp Leu Asp Cys Ser Ser Ser Asp Asp Glu Gly Ala Ala
290 295 300
Gln Asn Ser Thr Lys Pro Ser Ala Thr Lys Gly Thr Leu Gly Gly Met
305 310 315 320
Phe Gly Met Leu Lys Gly Leu Val Gly Ser Lys Ser Leu Ser Arg Glu
325 330 335
Asp Met Glu Ser Val Leu Asp Lys Met Arg Asp His Leu Ile Ala Lys
340 345 350
Asn Val Ala Ala Asp Ile Ala Val Gln Leu Cys Glu Ser Val Ala Asn
355 360 365
Lys Leu Glu Gly Lys Val Met Gly Thr Phe Ser Thr Val Thr Ser Thr
370 375 380
Val Lys Gln Ala Leu Gln Glu Ser Leu Val Gln Ile Leu Gln Pro Gln
385 390 395 400
Arg Arg Val Asp Met Leu Arg Asp Ile Met Asp Ala Gln Arg Arg Gln
405 410 415
Arg Pro Tyr Val Val Thr Phe Cys Gly Val Asn Gly Val Gly Lys Ser
420 425 430
Thr Asn Leu Ala Lys Ile Ser Phe Trp Leu Leu Glu Asn Gly Phe Ser
435 440 445
Val Leu Ile Ala Ala Cys Asp Thr Phe Arg Ala Gly Ala Val Glu Gln
450 455 460
Leu Arg Thr His Thr Arg Arg Leu Ser Ala Leu His Pro Pro Glu Lys
465 470 475 480
His Gly Gly Arg Thr Met Val Gln Leu Phe Glu Lys Gly Tyr Gly Lys
485 490 495
Asp Ala Ala Gly Ile Ala Met Glu Ala Ile Ala Phe Ala Arg Asn Gln
500 505 510
Gly Phe Asp Val Val Leu Val Asp Thr Ala Gly Arg Met Gln Asp Asn
515 520 525
Ala Pro Leu Met Thr Ala Leu Ala Lys Leu Ile Thr Val Asn Thr Pro
530 535 540
Asp Leu Val Leu Phe Val Gly Glu Ala Leu Val Gly Asn Glu Ala Val
545 550 555 560
Asp Gln Leu Val Lys Phe Asn Arg Ala Leu Ala Asp His Ser Met Ala
565 570 575
Gln Thr Pro Arg Leu Ile Asp Gly Ile Val Leu Thr Lys Phe Asp Thr
580 585 590
Ile Asp Asp Lys Val Gly Ala Ala Ile Ser Met Thr Tyr Ile Thr Ser
595 600 605
Lys Pro Ile Val Phe Val Gly Thr Gly Gln Thr Tyr Cys Asp Leu Arg
610 615 620
Ser Leu Asn Ala Lys Ala Val Val Ala Ala Leu Met Lys Ala
625 630 635
<210> 26
<211> 816
<212> ДНК
<213> Homo sapiens
<220>
<221> misc_feature
<223> hSRPR-бета
<400> 26
atggcttccg cggactcgcg ccgggtggca gatggcggcg gtgccggggg caccttccag 60
ccctacctag acaccttgcg gcaggagctg cagcagacgg acccaacgct gttgtcagta 120
gtggtggcgg ttcttgcggt gctgctgacg ctagtcttct ggaagttaat ccggagcaga 180
aggagcagtc agagagctgt tcttcttgtt ggcctttgtg attccgggaa aacgttgctc 240
tttgtcaggt tgttaacagg cctttataga gacactcaga cgtccattac tgacagctgt 300
gctgtataca gagtcaacaa taacaggggc aatagtctga ccttgattga ccttcccggc 360
catgagagtt tgaggcttca gttcttagag cggtttaagt cttcagccag ggctattgtg 420
tttgttgtgg atagtgcagc attccagcga gaggtgaaag atgtggctga gtttctgtat 480
caagtcctca ttgacagtat gggtctgaag aatacaccat cattcttaat agcctgcaat 540
aagcaagata ttgcaatggc aaaatcagca aagttaattc aacagcagct ggagaaagaa 600
ctcaacacct tacgagttac ccgttctgct gcccccagca cactggacag ttccagcact 660
gcccctgctc agctggggaa gaaaggcaaa gagtttgaat tctcacagtt gcccctcaaa 720
gtggagttcc tggagtgcag tgccaagggt ggaagagggg acgtgggctc tgctgacatc 780
caggacttgg agaaatggct ggctaaaatt gcctga 816
<210> 27
<211> 271
<212> ПРТ
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<223> hSRPR-бета
<400> 27
Met Ala Ser Ala Asp Ser Arg Arg Val Ala Asp Gly Gly Gly Ala Gly
1 5 10 15
Gly Thr Phe Gln Pro Tyr Leu Asp Thr Leu Arg Gln Glu Leu Gln Gln
20 25 30
Thr Asp Pro Thr Leu Leu Ser Val Val Val Ala Val Leu Ala Val Leu
35 40 45
Leu Thr Leu Val Phe Trp Lys Leu Ile Arg Ser Arg Arg Ser Ser Gln
50 55 60
Arg Ala Val Leu Leu Val Gly Leu Cys Asp Ser Gly Lys Thr Leu Leu
65 70 75 80
Phe Val Arg Leu Leu Thr Gly Leu Tyr Arg Asp Thr Gln Thr Ser Ile
85 90 95
Thr Asp Ser Cys Ala Val Tyr Arg Val Asn Asn Asn Arg Gly Asn Ser
100 105 110
Leu Thr Leu Ile Asp Leu Pro Gly His Glu Ser Leu Arg Leu Gln Phe
115 120 125
Leu Glu Arg Phe Lys Ser Ser Ala Arg Ala Ile Val Phe Val Val Asp
130 135 140
Ser Ala Ala Phe Gln Arg Glu Val Lys Asp Val Ala Glu Phe Leu Tyr
145 150 155 160
Gln Val Leu Ile Asp Ser Met Gly Leu Lys Asn Thr Pro Ser Phe Leu
165 170 175
Ile Ala Cys Asn Lys Gln Asp Ile Ala Met Ala Lys Ser Ala Lys Leu
180 185 190
Ile Gln Gln Gln Leu Glu Lys Glu Leu Asn Thr Leu Arg Val Thr Arg
195 200 205
Ser Ala Ala Pro Ser Thr Leu Asp Ser Ser Ser Thr Ala Pro Ala Gln
210 215 220
Leu Gly Lys Lys Gly Lys Glu Phe Glu Phe Ser Gln Leu Pro Leu Lys
225 230 235 240
Val Glu Phe Leu Glu Cys Ser Ala Lys Gly Gly Arg Gly Asp Val Gly
245 250 255
Ser Ala Asp Ile Gln Asp Leu Glu Lys Trp Leu Ala Lys Ile Ala
260 265 270
<210> 28
<211> 1779
<212> ДНК
<213> Homo sapiens
<220>
<221> misc_feature
<223> hCANX
<400> 28
atggaaggga agtggttgct gtgtatgtta ctggtgcttg gaactgctat tgttgaggct 60
catgatggac atgatgatga tgtgattgat attgaggatg accttgacga tgtcattgaa 120
gaggtagaag actcaaaacc agataccact gctcctcctt catctcccaa ggttacttac 180
aaagctccag ttccaacagg ggaagtatat tttgctgatt cttttgacag aggaactctg 240
tcagggtgga ttttatccaa agccaagaaa gacgataccg atgatgaaat tgccaaatat 300
gatggaaagt gggaggtaga ggaaatgaag gagtcaaagc ttccaggtga taaaggactt 360
gtgttgatgt ctcgggccaa gcatcatgcc atctctgcta aactgaacaa gcccttcctg 420
tttgacacca agcctctcat tgttcagtat gaggttaatt tccaaaatgg aatagaatgt 480
ggtggtgcct atgtgaaact gctttctaaa acaccagaac tcaacctgga tcagttccat 540
gacaagaccc cttatacgat tatgtttggt ccagataaat gtggagagga ctataaactg 600
cacttcatct tccgacacaa aaaccccaaa acgggtatct atgaagaaaa acatgctaag 660
aggccagatg cagatctgaa gacctatttt actgataaga aaacacatct ttacacacta 720
atcttgaatc cagataatag ttttgaaata ctggttgacc aatctgtggt gaatagtgga 780
aatctgctca atgacatgac tcctcctgta aatccttcac gtgaaattga ggacccagaa 840
gaccggaagc ccgaggattg ggatgaaaga ccaaaaatcc cagatccaga agctgtcaag 900
ccagatgact gggatgaaga tgcccctgct aagattccag atgaagaggc cacaaaaccc 960
gaaggctggt tagatgatga gcctgagtac gtacctgatc cagacgcaga gaaacctgag 1020
gattgggatg aagacatgga tggagaatgg gaggctcctc agattgccaa ccctagatgt 1080
gagtcagctc ctggatgtgg tgtctggcag cgacctgtga ttgacaaccc caattataaa 1140
ggcaaatgga agcctcctat gattgacaat cccagttacc agggaatctg gaaacccagg 1200
aaaataccaa atccagattt ctttgaagat ctggaacctt tcagaatgac tccttttagt 1260
gctattggtt tggagctgtg gtccatgacc tctgacattt tttttgacaa ctttatcatt 1320
tgtgctgatc gaagaatagt tgatgattgg gccaatgatg gatggggcct gaagaaagct 1380
gctgatgggg ctgctgagcc aggcgttgtg gggcagatga tcgaggcagc tgaagagcgc 1440
ccgtggctgt gggtagtcta tattctaact gtagcccttc ctgtgttcct ggttatcctc 1500
ttctgctgtt ctggaaagaa acagaccagt ggtatggagt ataagaaaac tgatgcacct 1560
caaccggatg tgaaggaaga ggaagaagag aaggaagagg aaaaggacaa gggagatgag 1620
gaggaggaag gagaagagaa acttgaagag aaacagaaaa gtgatgctga agaagatggt 1680
ggcactgtca gtcaagagga ggaagacaga aaacctaaag cagaggagga tgaaattttg 1740
aacagatcac caagaaacag aaagccacga agagagtga 1779
<210> 29
<211> 592
<212> ПРТ
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<223> hCANX
<400> 29
Met Glu Gly Lys Trp Leu Leu Cys Met Leu Leu Val Leu Gly Thr Ala
1 5 10 15
Ile Val Glu Ala His Asp Gly His Asp Asp Asp Val Ile Asp Ile Glu
20 25 30
Asp Asp Leu Asp Asp Val Ile Glu Glu Val Glu Asp Ser Lys Pro Asp
35 40 45
Thr Thr Ala Pro Pro Ser Ser Pro Lys Val Thr Tyr Lys Ala Pro Val
50 55 60
Pro Thr Gly Glu Val Tyr Phe Ala Asp Ser Phe Asp Arg Gly Thr Leu
65 70 75 80
Ser Gly Trp Ile Leu Ser Lys Ala Lys Lys Asp Asp Thr Asp Asp Glu
85 90 95
Ile Ala Lys Tyr Asp Gly Lys Trp Glu Val Glu Glu Met Lys Glu Ser
100 105 110
Lys Leu Pro Gly Asp Lys Gly Leu Val Leu Met Ser Arg Ala Lys His
115 120 125
His Ala Ile Ser Ala Lys Leu Asn Lys Pro Phe Leu Phe Asp Thr Lys
130 135 140
Pro Leu Ile Val Gln Tyr Glu Val Asn Phe Gln Asn Gly Ile Glu Cys
145 150 155 160
Gly Gly Ala Tyr Val Lys Leu Leu Ser Lys Thr Pro Glu Leu Asn Leu
165 170 175
Asp Gln Phe His Asp Lys Thr Pro Tyr Thr Ile Met Phe Gly Pro Asp
180 185 190
Lys Cys Gly Glu Asp Tyr Lys Leu His Phe Ile Phe Arg His Lys Asn
195 200 205
Pro Lys Thr Gly Ile Tyr Glu Glu Lys His Ala Lys Arg Pro Asp Ala
210 215 220
Asp Leu Lys Thr Tyr Phe Thr Asp Lys Lys Thr His Leu Tyr Thr Leu
225 230 235 240
Ile Leu Asn Pro Asp Asn Ser Phe Glu Ile Leu Val Asp Gln Ser Val
245 250 255
Val Asn Ser Gly Asn Leu Leu Asn Asp Met Thr Pro Pro Val Asn Pro
260 265 270
Ser Arg Glu Ile Glu Asp Pro Glu Asp Arg Lys Pro Glu Asp Trp Asp
275 280 285
Glu Arg Pro Lys Ile Pro Asp Pro Glu Ala Val Lys Pro Asp Asp Trp
290 295 300
Asp Glu Asp Ala Pro Ala Lys Ile Pro Asp Glu Glu Ala Thr Lys Pro
305 310 315 320
Glu Gly Trp Leu Asp Asp Glu Pro Glu Tyr Val Pro Asp Pro Asp Ala
325 330 335
Glu Lys Pro Glu Asp Trp Asp Glu Asp Met Asp Gly Glu Trp Glu Ala
340 345 350
Pro Gln Ile Ala Asn Pro Arg Cys Glu Ser Ala Pro Gly Cys Gly Val
355 360 365
Trp Gln Arg Pro Val Ile Asp Asn Pro Asn Tyr Lys Gly Lys Trp Lys
370 375 380
Pro Pro Met Ile Asp Asn Pro Ser Tyr Gln Gly Ile Trp Lys Pro Arg
385 390 395 400
Lys Ile Pro Asn Pro Asp Phe Phe Glu Asp Leu Glu Pro Phe Arg Met
405 410 415
Thr Pro Phe Ser Ala Ile Gly Leu Glu Leu Trp Ser Met Thr Ser Asp
420 425 430
Ile Phe Phe Asp Asn Phe Ile Ile Cys Ala Asp Arg Arg Ile Val Asp
435 440 445
Asp Trp Ala Asn Asp Gly Trp Gly Leu Lys Lys Ala Ala Asp Gly Ala
450 455 460
Ala Glu Pro Gly Val Val Gly Gln Met Ile Glu Ala Ala Glu Glu Arg
465 470 475 480
Pro Trp Leu Trp Val Val Tyr Ile Leu Thr Val Ala Leu Pro Val Phe
485 490 495
Leu Val Ile Leu Phe Cys Cys Ser Gly Lys Lys Gln Thr Ser Gly Met
500 505 510
Glu Tyr Lys Lys Thr Asp Ala Pro Gln Pro Asp Val Lys Glu Glu Glu
515 520 525
Glu Glu Lys Glu Glu Glu Lys Asp Lys Gly Asp Glu Glu Glu Glu Gly
530 535 540
Glu Glu Lys Leu Glu Glu Lys Gln Lys Ser Asp Ala Glu Glu Asp Gly
545 550 555 560
Gly Thr Val Ser Gln Glu Glu Glu Asp Arg Lys Pro Lys Ala Glu Glu
565 570 575
Asp Glu Ile Leu Asn Arg Ser Pro Arg Asn Arg Lys Pro Arg Arg Glu
580 585 590
<210> 30
<211> 972
<212> ДНК
<213> Homo sapiens
<220>
<221> misc_feature
<223> hUCP4
<400> 30
atgtccgtcc cggaggagga ggagaggctt ttgccgctga cccagagatg gccccgagcg 60
agcaaattcc tactgtccgg ctgcgcggct accgtggccg agctagcaac ctttcccctg 120
gatctcacaa aaactcgact ccaaatgcaa ggagaagcag ctcttgctcg gttgggagac 180
ggtgcaagag aatctgcccc ctatagggga atggtgcgca cagccctagg gatcattgaa 240
gaggaaggct ttctaaagct ttggcaagga gtgacacccg ccatttacag acacgtagtg 300
tattctggag gtcgaatggt cacatatgaa catctccgag aggttgtgtt tggcaaaagt 360
gaagatgagc attatcccct ttggaaatca gtcattggag ggatgatggc tggtgttatt 420
ggccagtttt tagccaatcc aactgaccta gtgaaggttc agatgcaaat ggaaggaaaa 480
aggaaactgg aaggaaaacc attgcgattt cgtggtgtac atcatgcatt tgcaaaaatc 540
ttagctgaag gaggaatacg agggctttgg gcaggctggg tacccaatat acaaagagca 600
gcactggtga atatgggaga tttaaccact tatgatacag tgaaacacta cttggtattg 660
aatacaccac ttgaggacaa tatcatgact cacggtttat caagtttatg ttctggactg 720
gtagcttcta ttctgggaac accagccgat gtcatcaaaa gcagaataat gaatcaacca 780
cgagataaac aaggaagggg acttttgtat aaatcatcga ctgactgctt gattcaggct 840
gttcaaggtg aaggattcat gagtctatat aaaggctttt taccatcttg gctgagaatg 900
accccttggt caatggtgtt ctggcttact tatgaaaaaa tcagagagat gagtggagtc 960
agtccatttt aa 972
<210> 31
<211> 323
<212> ПРТ
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<223> hUCP4
<400> 31
Met Ser Val Pro Glu Glu Glu Glu Arg Leu Leu Pro Leu Thr Gln Arg
1 5 10 15
Trp Pro Arg Ala Ser Lys Phe Leu Leu Ser Gly Cys Ala Ala Thr Val
20 25 30
Ala Glu Leu Ala Thr Phe Pro Leu Asp Leu Thr Lys Thr Arg Leu Gln
35 40 45
Met Gln Gly Glu Ala Ala Leu Ala Arg Leu Gly Asp Gly Ala Arg Glu
50 55 60
Ser Ala Pro Tyr Arg Gly Met Val Arg Thr Ala Leu Gly Ile Ile Glu
65 70 75 80
Glu Glu Gly Phe Leu Lys Leu Trp Gln Gly Val Thr Pro Ala Ile Tyr
85 90 95
Arg His Val Val Tyr Ser Gly Gly Arg Met Val Thr Tyr Glu His Leu
100 105 110
Arg Glu Val Val Phe Gly Lys Ser Glu Asp Glu His Tyr Pro Leu Trp
115 120 125
Lys Ser Val Ile Gly Gly Met Met Ala Gly Val Ile Gly Gln Phe Leu
130 135 140
Ala Asn Pro Thr Asp Leu Val Lys Val Gln Met Gln Met Glu Gly Lys
145 150 155 160
Arg Lys Leu Glu Gly Lys Pro Leu Arg Phe Arg Gly Val His His Ala
165 170 175
Phe Ala Lys Ile Leu Ala Glu Gly Gly Ile Arg Gly Leu Trp Ala Gly
180 185 190
Trp Val Pro Asn Ile Gln Arg Ala Ala Leu Val Asn Met Gly Asp Leu
195 200 205
Thr Thr Tyr Asp Thr Val Lys His Tyr Leu Val Leu Asn Thr Pro Leu
210 215 220
Glu Asp Asn Ile Met Thr His Gly Leu Ser Ser Leu Cys Ser Gly Leu
225 230 235 240
Val Ala Ser Ile Leu Gly Thr Pro Ala Asp Val Ile Lys Ser Arg Ile
245 250 255
Met Asn Gln Pro Arg Asp Lys Gln Gly Arg Gly Leu Leu Tyr Lys Ser
260 265 270
Ser Thr Asp Cys Leu Ile Gln Ala Val Gln Gly Glu Gly Phe Met Ser
275 280 285
Leu Tyr Lys Gly Phe Leu Pro Ser Trp Leu Arg Met Thr Pro Trp Ser
290 295 300
Met Val Phe Trp Leu Thr Tyr Glu Lys Ile Arg Glu Met Ser Gly Val
305 310 315 320
Ser Pro Phe
<210> 32
<211> 1014
<212> ДНК
<213> Homo sapiens
<220>
<221> misc_feature
<223> hCMPSAT
<400> 32
atggctgccc cgagagacaa tgtcacttta ttattcaagt tatactgctt ggcagtgatg 60
accctgatgg ctgcagtcta taccatagct ttaagataca caaggacatc agacaaagaa 120
ctctactttt caaccacagc cgtgtgtatc acagaagtta taaagttatt gctaagtgtg 180
ggaattttag ctaaagaaac tggtagtctg ggtagattca aagcatcttt aagagaaaat 240
gtcttgggga gccccaagga actgttgaag ttaagtgtgc catcgttagt gtatgctgtt 300
cagaacaaca tggctttcct agctcttagc aatctggatg cagcagtgta ccaggtgacc 360
taccagttga agattccgtg tactgcttta tgcactgttt taatgttaaa ccggacactc 420
agcaaattac agtgggtttc agtttttatg ctgtgtgctg gagttacgct tgtacagtgg 480
aaaccagccc aagctacaaa agtggtggtg gaacaaaatc cattattagg gtttggcgct 540
atagctattg ctgtattgtg ctcaggattt gcaggagtat attttgaaaa agttttaaag 600
agttcagata cttctctttg ggtgagaaac attcaaatgt atctatcagg gattattgtg 660
acattagctg gcgtctactt gtcagatgga gctgaaatta aagaaaaagg atttttctat 720
ggttacacat attatgtctg gtttgtcatc tttcttgcaa gtgttggtgg cctctacact 780
tctgttgtgg ttaagtacac agacaacatc atgaaaggct tttctgcagc agcggccatt 840
gtcctttcca ccattgcttc agtaatgctg tttggattac agataacact cacctttgcc 900
ctgggtactc ttcttgtatg tgtttccata tatctctatg gattacccag acaagacact 960
acatccatcc aacaaggaga aacagcttca aaggagagag ttattggtgt gtga 1014
<210> 33
<211> 337
<212> ПРТ
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<223> hCMPSAT
<400> 33
Met Ala Ala Pro Arg Asp Asn Val Thr Leu Leu Phe Lys Leu Tyr Cys
1 5 10 15
Leu Ala Val Met Thr Leu Met Ala Ala Val Tyr Thr Ile Ala Leu Arg
20 25 30
Tyr Thr Arg Thr Ser Asp Lys Glu Leu Tyr Phe Ser Thr Thr Ala Val
35 40 45
Cys Ile Thr Glu Val Ile Lys Leu Leu Leu Ser Val Gly Ile Leu Ala
50 55 60
Lys Glu Thr Gly Ser Leu Gly Arg Phe Lys Ala Ser Leu Arg Glu Asn
65 70 75 80
Val Leu Gly Ser Pro Lys Glu Leu Leu Lys Leu Ser Val Pro Ser Leu
85 90 95
Val Tyr Ala Val Gln Asn Asn Met Ala Phe Leu Ala Leu Ser Asn Leu
100 105 110
Asp Ala Ala Val Tyr Gln Val Thr Tyr Gln Leu Lys Ile Pro Cys Thr
115 120 125
Ala Leu Cys Thr Val Leu Met Leu Asn Arg Thr Leu Ser Lys Leu Gln
130 135 140
Trp Val Ser Val Phe Met Leu Cys Ala Gly Val Thr Leu Val Gln Trp
145 150 155 160
Lys Pro Ala Gln Ala Thr Lys Val Val Val Glu Gln Asn Pro Leu Leu
165 170 175
Gly Phe Gly Ala Ile Ala Ile Ala Val Leu Cys Ser Gly Phe Ala Gly
180 185 190
Val Tyr Phe Glu Lys Val Leu Lys Ser Ser Asp Thr Ser Leu Trp Val
195 200 205
Arg Asn Ile Gln Met Tyr Leu Ser Gly Ile Ile Val Thr Leu Ala Gly
210 215 220
Val Tyr Leu Ser Asp Gly Ala Glu Ile Lys Glu Lys Gly Phe Phe Tyr
225 230 235 240
Gly Tyr Thr Tyr Tyr Val Trp Phe Val Ile Phe Leu Ala Ser Val Gly
245 250 255
Gly Leu Tyr Thr Ser Val Val Val Lys Tyr Thr Asp Asn Ile Met Lys
260 265 270
Gly Phe Ser Ala Ala Ala Ala Ile Val Leu Ser Thr Ile Ala Ser Val
275 280 285
Met Leu Phe Gly Leu Gln Ile Thr Leu Thr Phe Ala Leu Gly Thr Leu
290 295 300
Leu Val Cys Val Ser Ile Tyr Leu Tyr Gly Leu Pro Arg Gln Asp Thr
305 310 315 320
Thr Ser Ile Gln Gln Gly Glu Thr Ala Ser Lys Glu Arg Val Ile Gly
325 330 335
Val
<210> 34
<211> 1212
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Оптимизированная последовательность
<220>
<221> misc_feature
<223> rST6Gal1
<400> 34
atgatccaca ccaacctgaa gaagaagttc tccctgttca tcctggtgtt tctgctgttc 60
gccgtgatct gcgtgtggaa gaagggctcc gactacgagg ctctgaccct gcaggccaaa 120
gaattccaga tgcccaagtc ccaagaaaag gtggcaatgg gctccgccag ccaggtggtg 180
ttctccaact ccaagcagga ccctaaagag gacatcccta tcctgtccta ccacagagtg 240
accgccaaag tgaagcctca gcctagcttc caggtctggg acaaggactc cacctacagc 300
aagctgaacc ctcggctgct gaagatctgg cggaactacc tgaacatgaa caagtacaag 360
gtgtcctaca agggccctgg ccctggcgtg aagttctccg tggaggccct gaggtgccac 420
ctgagggacc acgtgaacgt gtccatgatc gaggccaccg acttcccatt caacaccacc 480
gagtgggagg gctacctgcc taaagagaac ttccggacca aagtgggccc ttggcagaga 540
tgcgccgtgg tgtcctccgc cggctccctg aagaactctc agctgggccg ggagatcgac 600
aaccacgacg ccgtgctgag gttcaacggc gctcctaccg acaacttcca gcaggacgtg 660
ggctccaaga ccaccatcag actgatgaac agccagctgg tgaccaccga gaagcggttt 720
ctgaaggact ccctgtacac cgagggcatc ctgatcgtgt gggacccttc cgtgtaccac 780
gccgacatcc ctaagtggta tcagaagccc gactacaact tcttcgagac atacaagtcc 840
taccggcggc tgaacccttc ccagcctttc tacatcctga agccccagat gccttgggag 900
ctgtgggaca tcatccagga aatctccgcc gacctgatcc agcctaaccc tccttcctcc 960
ggcatgctgg gcatcatcat catgatgacc ctgtgcgacc aggtggacat ctacgagttt 1020
ctgccttcca agagaaagac cgacgtgtgc tactaccacc agaagttctt cgactccgcc 1080
tgcacaatgg gcgcctacca ccccctgctg ttcgagaaga acatggtgaa gcacctgaac 1140
gagggcaccg acgaggacat ctacctgttc ggcaaggcca ccctgtccgg cttccggaac 1200
atccggtgct ga 1212
<210> 35
<211> 403
<212> ПРТ
<213> Rattus norvegicus
<220>
<221> MISC_FEATURE
<223> rST6Gal1
<400> 35
Met Ile His Thr Asn Leu Lys Lys Lys Phe Ser Leu Phe Ile Leu Val
1 5 10 15
Phe Leu Leu Phe Ala Val Ile Cys Val Trp Lys Lys Gly Ser Asp Tyr
20 25 30
Glu Ala Leu Thr Leu Gln Ala Lys Glu Phe Gln Met Pro Lys Ser Gln
35 40 45
Glu Lys Val Ala Met Gly Ser Ala Ser Gln Val Val Phe Ser Asn Ser
50 55 60
Lys Gln Asp Pro Lys Glu Asp Ile Pro Ile Leu Ser Tyr His Arg Val
65 70 75 80
Thr Ala Lys Val Lys Pro Gln Pro Ser Phe Gln Val Trp Asp Lys Asp
85 90 95
Ser Thr Tyr Ser Lys Leu Asn Pro Arg Leu Leu Lys Ile Trp Arg Asn
100 105 110
Tyr Leu Asn Met Asn Lys Tyr Lys Val Ser Tyr Lys Gly Pro Gly Pro
115 120 125
Gly Val Lys Phe Ser Val Glu Ala Leu Arg Cys His Leu Arg Asp His
130 135 140
Val Asn Val Ser Met Ile Glu Ala Thr Asp Phe Pro Phe Asn Thr Thr
145 150 155 160
Glu Trp Glu Gly Tyr Leu Pro Lys Glu Asn Phe Arg Thr Lys Val Gly
165 170 175
Pro Trp Gln Arg Cys Ala Val Val Ser Ser Ala Gly Ser Leu Lys Asn
180 185 190
Ser Gln Leu Gly Arg Glu Ile Asp Asn His Asp Ala Val Leu Arg Phe
195 200 205
Asn Gly Ala Pro Thr Asp Asn Phe Gln Gln Asp Val Gly Ser Lys Thr
210 215 220
Thr Ile Arg Leu Met Asn Ser Gln Leu Val Thr Thr Glu Lys Arg Phe
225 230 235 240
Leu Lys Asp Ser Leu Tyr Thr Glu Gly Ile Leu Ile Val Trp Asp Pro
245 250 255
Ser Val Tyr His Ala Asp Ile Pro Lys Trp Tyr Gln Lys Pro Asp Tyr
260 265 270
Asn Phe Phe Glu Thr Tyr Lys Ser Tyr Arg Arg Leu Asn Pro Ser Gln
275 280 285
Pro Phe Tyr Ile Leu Lys Pro Gln Met Pro Trp Glu Leu Trp Asp Ile
290 295 300
Ile Gln Glu Ile Ser Ala Asp Leu Ile Gln Pro Asn Pro Pro Ser Ser
305 310 315 320
Gly Met Leu Gly Ile Ile Ile Met Met Thr Leu Cys Asp Gln Val Asp
325 330 335
Ile Tyr Glu Phe Leu Pro Ser Lys Arg Lys Thr Asp Val Cys Tyr Tyr
340 345 350
His Gln Lys Phe Phe Asp Ser Ala Cys Thr Met Gly Ala Tyr His Pro
355 360 365
Leu Leu Phe Glu Lys Asn Met Val Lys His Leu Asn Glu Gly Thr Asp
370 375 380
Glu Asp Ile Tyr Leu Phe Gly Lys Ala Thr Leu Ser Gly Phe Arg Asn
385 390 395 400
Ile Arg Cys
<210> 36
<211> 957
<212> ДНК
<213> Homo sapiens
<220>
<221> misc_feature
<223> hCOMSC
<400> 36
atgctttctg aaagcagctc ctttttgaag ggtgtgatgc ttggaagcat tttctgtgct 60
ttgatcacta tgctaggaca cattaggatt ggtcatggaa atagaatgca ccaccatgag 120
catcatcacc tacaagctcc taacaaagaa gatatcttga aaatttcaga ggatgagcgc 180
atggagctca gtaagagctt tcgagtatac tgtattatcc ttgtaaaacc caaagatgtg 240
agtctttggg ctgcagtaaa ggagacttgg accaaacact gtgacaaagc agagttcttc 300
agttctgaaa atgttaaagt gtttgagtca attaatatgg acacaaatga catgtggtta 360
atgatgagaa aagcttacaa atacgccttt gataagtata gagaccaata caactggttc 420
ttccttgcac gccccactac gtttgctatc attgaaaacc taaagtattt tttgttaaaa 480
aaggatccat cacagccttt ctatctaggc cacactataa aatctggaga ccttgaatat 540
gtgggtatgg aaggaggaat tgtcttaagt gtagaatcaa tgaaaagact taacagcctt 600
ctcaatatcc cagaaaagtg tcctgaacag ggagggatga tttggaagat atctgaagat 660
aaacagctag cagtttgcct gaaatatgct ggagtatttg cagaaaatgc agaagatgct 720
gatggaaaag atgtatttaa taccaaatct gttgggcttt ctattaaaga ggcaatgact 780
tatcacccca accaggtagt agaaggctgt tgttcagata tggctgttac ttttaatgga 840
ctgactccaa atcagatgca tgtgatgatg tatggggtat accgccttag ggcatttggg 900
catattttca atgatgcatt ggttttctta cctccaaatg gttctgacaa tgactga 957
<210> 37
<211> 318
<212> ПРТ
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<223> hCOMSC
<400> 37
Met Leu Ser Glu Ser Ser Ser Phe Leu Lys Gly Val Met Leu Gly Ser
1 5 10 15
Ile Phe Cys Ala Leu Ile Thr Met Leu Gly His Ile Arg Ile Gly His
20 25 30
Gly Asn Arg Met His His His Glu His His His Leu Gln Ala Pro Asn
35 40 45
Lys Glu Asp Ile Leu Lys Ile Ser Glu Asp Glu Arg Met Glu Leu Ser
50 55 60
Lys Ser Phe Arg Val Tyr Cys Ile Ile Leu Val Lys Pro Lys Asp Val
65 70 75 80
Ser Leu Trp Ala Ala Val Lys Glu Thr Trp Thr Lys His Cys Asp Lys
85 90 95
Ala Glu Phe Phe Ser Ser Glu Asn Val Lys Val Phe Glu Ser Ile Asn
100 105 110
Met Asp Thr Asn Asp Met Trp Leu Met Met Arg Lys Ala Tyr Lys Tyr
115 120 125
Ala Phe Asp Lys Tyr Arg Asp Gln Tyr Asn Trp Phe Phe Leu Ala Arg
130 135 140
Pro Thr Thr Phe Ala Ile Ile Glu Asn Leu Lys Tyr Phe Leu Leu Lys
145 150 155 160
Lys Asp Pro Ser Gln Pro Phe Tyr Leu Gly His Thr Ile Lys Ser Gly
165 170 175
Asp Leu Glu Tyr Val Gly Met Glu Gly Gly Ile Val Leu Ser Val Glu
180 185 190
Ser Met Lys Arg Leu Asn Ser Leu Leu Asn Ile Pro Glu Lys Cys Pro
195 200 205
Glu Gln Gly Gly Met Ile Trp Lys Ile Ser Glu Asp Lys Gln Leu Ala
210 215 220
Val Cys Leu Lys Tyr Ala Gly Val Phe Ala Glu Asn Ala Glu Asp Ala
225 230 235 240
Asp Gly Lys Asp Val Phe Asn Thr Lys Ser Val Gly Leu Ser Ile Lys
245 250 255
Glu Ala Met Thr Tyr His Pro Asn Gln Val Val Glu Gly Cys Cys Ser
260 265 270
Asp Met Ala Val Thr Phe Asn Gly Leu Thr Pro Asn Gln Met His Val
275 280 285
Met Met Tyr Gly Val Tyr Arg Leu Arg Ala Phe Gly His Ile Phe Asn
290 295 300
Asp Ala Leu Val Phe Leu Pro Pro Asn Gly Ser Asp Asn Asp
305 310 315
<210> 38
<211> 1092
<212> ДНК
<213> Homo sapiens
<220>
<221> misc_feature
<223> hT-синтаза
<400> 38
atggcctcta aatcctggct gaatttttta accttcctct gtggatcagc aataggattt 60
cttttatgtt ctcagctatt tagtattttg ttgggagaaa aggttgacac ccagcctaat 120
gttcttcata atgatcctca tgcaaggcat tcagatgata atggacagaa tcatctagaa 180
ggacaaatga acttcaatgc agattctagc caacataaag atgagaacac agacattgct 240
gaaaacctct atcagaaagt tagaattctt tgctgggtta tgaccggccc tcaaaaccta 300
gagaaaaagg ccaaacacgt caaagctact tgggcccagc gttgtaacaa agtgttgttt 360
atgagttcag aagaaaataa agacttccct gctgtgggac tgaaaaccaa agaaggcaga 420
gatcaactat actggaaaac aattaaagct tttcagtatg ttcatgaaca ttatttagaa 480
gatgctgatt ggtttttgaa agcagatgat gacacgtatg tcatactaga caatttgagg 540
tggcttcttt caaaatacga ccctgaagaa cccatttact ttgggagaag atttaagcct 600
tatgtaaagc agggctacat gagtggagga gcaggatatg tactaagcaa agaagccttg 660
aaaagatttg ttgatgcatt taaaacagac aagtgtacac atagttcctc cattgaagac 720
ttagcactgg ggagatgcat ggaaattatg aatgtagaag caggagattc cagagatacc 780
attggaaaag aaacttttca tccctttgtg ccagaacacc atttaattaa aggttatcta 840
cctagaacgt tttggtactg gaattacaac tattatcctc ctgtagaggg tcctggttgc 900
tgctctgatc ttgcagtttc ttttcactat gttgattcta caaccatgta tgagttagaa 960
tacctcgttt atcatcttcg tccatatggt tatttataca gatatcaacc taccttacct 1020
gaacgtatac taaaggaaat tagtcaagca aacaaaaatg aagatacaaa agtgaagtta 1080
ggaaatcctt ga 1092
<210> 39
<211> 363
<212> ПРТ
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<223> hT-синтаза
<400> 39
Met Ala Ser Lys Ser Trp Leu Asn Phe Leu Thr Phe Leu Cys Gly Ser
1 5 10 15
Ala Ile Gly Phe Leu Leu Cys Ser Gln Leu Phe Ser Ile Leu Leu Gly
20 25 30
Glu Lys Val Asp Thr Gln Pro Asn Val Leu His Asn Asp Pro His Ala
35 40 45
Arg His Ser Asp Asp Asn Gly Gln Asn His Leu Glu Gly Gln Met Asn
50 55 60
Phe Asn Ala Asp Ser Ser Gln His Lys Asp Glu Asn Thr Asp Ile Ala
65 70 75 80
Glu Asn Leu Tyr Gln Lys Val Arg Ile Leu Cys Trp Val Met Thr Gly
85 90 95
Pro Gln Asn Leu Glu Lys Lys Ala Lys His Val Lys Ala Thr Trp Ala
100 105 110
Gln Arg Cys Asn Lys Val Leu Phe Met Ser Ser Glu Glu Asn Lys Asp
115 120 125
Phe Pro Ala Val Gly Leu Lys Thr Lys Glu Gly Arg Asp Gln Leu Tyr
130 135 140
Trp Lys Thr Ile Lys Ala Phe Gln Tyr Val His Glu His Tyr Leu Glu
145 150 155 160
Asp Ala Asp Trp Phe Leu Lys Ala Asp Asp Asp Thr Tyr Val Ile Leu
165 170 175
Asp Asn Leu Arg Trp Leu Leu Ser Lys Tyr Asp Pro Glu Glu Pro Ile
180 185 190
Tyr Phe Gly Arg Arg Phe Lys Pro Tyr Val Lys Gln Gly Tyr Met Ser
195 200 205
Gly Gly Ala Gly Tyr Val Leu Ser Lys Glu Ala Leu Lys Arg Phe Val
210 215 220
Asp Ala Phe Lys Thr Asp Lys Cys Thr His Ser Ser Ser Ile Glu Asp
225 230 235 240
Leu Ala Leu Gly Arg Cys Met Glu Ile Met Asn Val Glu Ala Gly Asp
245 250 255
Ser Arg Asp Thr Ile Gly Lys Glu Thr Phe His Pro Phe Val Pro Glu
260 265 270
His His Leu Ile Lys Gly Tyr Leu Pro Arg Thr Phe Trp Tyr Trp Asn
275 280 285
Tyr Asn Tyr Tyr Pro Pro Val Glu Gly Pro Gly Cys Cys Ser Asp Leu
290 295 300
Ala Val Ser Phe His Tyr Val Asp Ser Thr Thr Met Tyr Glu Leu Glu
305 310 315 320
Tyr Leu Val Tyr His Leu Arg Pro Tyr Gly Tyr Leu Tyr Arg Tyr Gln
325 330 335
Pro Thr Leu Pro Glu Arg Ile Leu Lys Glu Ile Ser Gln Ala Asn Lys
340 345 350
Asn Glu Asp Thr Lys Val Lys Leu Gly Asn Pro
355 360
<210> 40
<211> 1605
<212> ДНК
<213> Homo sapiens
<220>
<221> misc_feature
<223> hP4HA1
<400> 40
atgatctggt atatattaat tataggaatt ctgcttcccc agtctttggc tcatccaggc 60
ttttttactt caattggtca gatgactgat ttgatccata ctgagaaaga tctggtgact 120
tctctgaaag attatattaa ggcagaagag gacaagttag aacaaataaa aaaatgggca 180
gagaagttag atcggctaac tagtacagcg acaaaagatc cagaaggatt tgttgggcat 240
ccagtaaatg cattcaaatt aatgaaacgt ctgaatactg agtggagtga gttggagaat 300
ctggtcctta aggatatgtc agatggcttt atctctaacc taaccattca gagacagtac 360
tttcctaatg atgaagatca ggttggggca gccaaagctc tgttacgtct ccaggatacc 420
tacaatttgg atacagatac catctcaaag ggtaatcttc caggagtgaa acacaaatct 480
tttctaacgg ctgaggactg ctttgagttg ggcaaagtgg cctatacaga agcagattat 540
taccatacgg aactgtggat ggaacaagcc ctaaggcaac tggatgaagg cgagatttct 600
accatagata aagtctctgt tctagattat ttgagctatg cggtatatca gcagggagac 660
ctggataagg cacttttgct cacaaagaag cttcttgaac tagatcctga acatcagaga 720
gctaatggta acttaaaata ttttgagtat ataatggcta aagaaaaaga tgtcaataag 780
tctgcttcag atgaccaatc tgatcagaaa actacaccaa agaaaaaagg ggttgctgtg 840
gattacctgc cagagagaca gaagtacgaa atgctgtgcc gtggggaggg tatcaaaatg 900
acccctcgga gacagaaaaa actcttttgc cgctaccatg atggaaaccg taatcctaaa 960
tttattctgg ctccagctaa acaggaggat gaatgggaca agcctcgtat tattcgcttc 1020
catgatatta tttctgatgc agaaattgaa atcgtcaaag acctagcaaa accaaggctg 1080
agccgagcta cagtacatga ccctgagact ggaaaattga ccacagcaca gtacagagta 1140
tctaagagtg cctggctctc tggctatgaa aatcctgtgg tgtctcgaat taatatgaga 1200
atacaagatc taacaggact agatgtttcc acagcagagg aattacaggt agcaaattat 1260
ggagttggag gacagtatga accccatttt gactttgcac ggaaagatga gccagatgct 1320
ttcaaagagc tggggacagg aaatagaatt gctacatggc tgttttatat gagtgatgtg 1380
tctgcaggag gagccactgt ttttcctgaa gttggagcta gtgtttggcc caaaaaagga 1440
actgctgttt tctggtataa tctgtttgcc agtggagaag gagattatag tacacggcat 1500
gcagcctgtc cagtgctagt tggcaacaaa tgggtatcca ataaatggct ccatgaacgt 1560
ggacaagaat ttcgaagacc ttgtacgttg tcagaattgg aatga 1605
<210> 41
<211> 534
<212> ПРТ
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<223> hP4HA1
<400> 41
Met Ile Trp Tyr Ile Leu Ile Ile Gly Ile Leu Leu Pro Gln Ser Leu
1 5 10 15
Ala His Pro Gly Phe Phe Thr Ser Ile Gly Gln Met Thr Asp Leu Ile
20 25 30
His Thr Glu Lys Asp Leu Val Thr Ser Leu Lys Asp Tyr Ile Lys Ala
35 40 45
Glu Glu Asp Lys Leu Glu Gln Ile Lys Lys Trp Ala Glu Lys Leu Asp
50 55 60
Arg Leu Thr Ser Thr Ala Thr Lys Asp Pro Glu Gly Phe Val Gly His
65 70 75 80
Pro Val Asn Ala Phe Lys Leu Met Lys Arg Leu Asn Thr Glu Trp Ser
85 90 95
Glu Leu Glu Asn Leu Val Leu Lys Asp Met Ser Asp Gly Phe Ile Ser
100 105 110
Asn Leu Thr Ile Gln Arg Gln Tyr Phe Pro Asn Asp Glu Asp Gln Val
115 120 125
Gly Ala Ala Lys Ala Leu Leu Arg Leu Gln Asp Thr Tyr Asn Leu Asp
130 135 140
Thr Asp Thr Ile Ser Lys Gly Asn Leu Pro Gly Val Lys His Lys Ser
145 150 155 160
Phe Leu Thr Ala Glu Asp Cys Phe Glu Leu Gly Lys Val Ala Tyr Thr
165 170 175
Glu Ala Asp Tyr Tyr His Thr Glu Leu Trp Met Glu Gln Ala Leu Arg
180 185 190
Gln Leu Asp Glu Gly Glu Ile Ser Thr Ile Asp Lys Val Ser Val Leu
195 200 205
Asp Tyr Leu Ser Tyr Ala Val Tyr Gln Gln Gly Asp Leu Asp Lys Ala
210 215 220
Leu Leu Leu Thr Lys Lys Leu Leu Glu Leu Asp Pro Glu His Gln Arg
225 230 235 240
Ala Asn Gly Asn Leu Lys Tyr Phe Glu Tyr Ile Met Ala Lys Glu Lys
245 250 255
Asp Val Asn Lys Ser Ala Ser Asp Asp Gln Ser Asp Gln Lys Thr Thr
260 265 270
Pro Lys Lys Lys Gly Val Ala Val Asp Tyr Leu Pro Glu Arg Gln Lys
275 280 285
Tyr Glu Met Leu Cys Arg Gly Glu Gly Ile Lys Met Thr Pro Arg Arg
290 295 300
Gln Lys Lys Leu Phe Cys Arg Tyr His Asp Gly Asn Arg Asn Pro Lys
305 310 315 320
Phe Ile Leu Ala Pro Ala Lys Gln Glu Asp Glu Trp Asp Lys Pro Arg
325 330 335
Ile Ile Arg Phe His Asp Ile Ile Ser Asp Ala Glu Ile Glu Ile Val
340 345 350
Lys Asp Leu Ala Lys Pro Arg Leu Ser Arg Ala Thr Val His Asp Pro
355 360 365
Glu Thr Gly Lys Leu Thr Thr Ala Gln Tyr Arg Val Ser Lys Ser Ala
370 375 380
Trp Leu Ser Gly Tyr Glu Asn Pro Val Val Ser Arg Ile Asn Met Arg
385 390 395 400
Ile Gln Asp Leu Thr Gly Leu Asp Val Ser Thr Ala Glu Glu Leu Gln
405 410 415
Val Ala Asn Tyr Gly Val Gly Gly Gln Tyr Glu Pro His Phe Asp Phe
420 425 430
Ala Arg Lys Asp Glu Pro Asp Ala Phe Lys Glu Leu Gly Thr Gly Asn
435 440 445
Arg Ile Ala Thr Trp Leu Phe Tyr Met Ser Asp Val Ser Ala Gly Gly
450 455 460
Ala Thr Val Phe Pro Glu Val Gly Ala Ser Val Trp Pro Lys Lys Gly
465 470 475 480
Thr Ala Val Phe Trp Tyr Asn Leu Phe Ala Ser Gly Glu Gly Asp Tyr
485 490 495
Ser Thr Arg His Ala Ala Cys Pro Val Leu Val Gly Asn Lys Trp Val
500 505 510
Ser Asn Lys Trp Leu His Glu Arg Gly Gln Glu Phe Arg Arg Pro Cys
515 520 525
Thr Leu Ser Glu Leu Glu
530
<210> 42
<211> 1527
<212> ДНК
<213> Homo sapiens
<220>
<221> misc_feature
<223> hP4HB
<400> 42
atgctgcgcc gcgctctgct gtgcctggcc gtggccgccc tggtgcgcgc cgacgccccc 60
gaggaggagg accacgtcct ggtgctgcgg aaaagcaact tcgcggaggc gctggcggcc 120
cacaagtacc tgctggtgga gttctatgcc ccttggtgtg gccactgcaa ggctctggcc 180
cctgagtatg ccaaagccgc tgggaagctg aaggcagaag gttccgagat caggttggcc 240
aaggtggacg ccacggagga gtctgacctg gcccagcagt acggcgtgcg cggctatccc 300
accatcaagt tcttcaggaa tggagacacg gcttccccca aggaatatac agctggcaga 360
gaggctgatg acatcgtgaa ctggctgaag aagcgcacgg gcccggctgc caccaccctg 420
cctgacggcg cagctgcaga gtccttggtg gagtccagcg aggtggctgt catcggcttc 480
ttcaaggacg tggagtcgga ctctgccaag cagtttttgc aggcagcaga ggccatcgat 540
gacataccat ttgggatcac ttccaacagt gacgtgttct ccaaatacca gctcgacaaa 600
gatggggttg tcctctttaa gaagtttgat gaaggccgga acaactttga aggggaggtc 660
accaaggaga acctgctgga ctttatcaaa cacaaccagc tgccccttgt catcgagttc 720
accgagcaga cagccccgaa gatttttgga ggtgaaatca agactcacat cctgctgttc 780
ttgcccaaga gtgtgtctga ctatgacggc aaactgagca acttcaaaac agcagccgag 840
agcttcaagg gcaagatcct gttcatcttc atcgacagcg accacaccga caaccagcgc 900
atcctcgagt tctttggcct gaagaaggaa gagtgcccgg ccgtgcgcct catcaccctg 960
gaggaggaga tgaccaagta caagcccgaa tcggaggagc tgacggcaga gaggatcaca 1020
gagttctgcc accgcttcct ggagggcaaa atcaagcccc acctgatgag ccaggagctg 1080
ccggaggact gggacaagca gcctgtcaag gtgcttgttg ggaagaactt tgaagacgtg 1140
gcttttgatg agaaaaaaaa cgtctttgtg gagttctatg ccccatggtg tggtcactgc 1200
aaacagttgg ctcccatttg ggataaactg ggagagacgt acaaggacca tgagaacatc 1260
gtcatcgcca agatggactc gactgccaac gaggtggagg ccgtcaaagt gcacagcttc 1320
cccacactca agttctttcc tgccagtgcc gacaggacgg tcattgatta caacggggaa 1380
cgcacgctgg atggttttaa gaaattcctg gagagcggtg gccaggatgg ggcaggggat 1440
gatgacgatc tcgaggacct ggaagaagca gaggagccag acatggagga agacgatgat 1500
cagaaagctg tgaaagatga actgtaa 1527
<210> 43
<211> 508
<212> ПРТ
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<223> hP4HB
<400> 43
Met Leu Arg Arg Ala Leu Leu Cys Leu Ala Val Ala Ala Leu Val Arg
1 5 10 15
Ala Asp Ala Pro Glu Glu Glu Asp His Val Leu Val Leu Arg Lys Ser
20 25 30
Asn Phe Ala Glu Ala Leu Ala Ala His Lys Tyr Leu Leu Val Glu Phe
35 40 45
Tyr Ala Pro Trp Cys Gly His Cys Lys Ala Leu Ala Pro Glu Tyr Ala
50 55 60
Lys Ala Ala Gly Lys Leu Lys Ala Glu Gly Ser Glu Ile Arg Leu Ala
65 70 75 80
Lys Val Asp Ala Thr Glu Glu Ser Asp Leu Ala Gln Gln Tyr Gly Val
85 90 95
Arg Gly Tyr Pro Thr Ile Lys Phe Phe Arg Asn Gly Asp Thr Ala Ser
100 105 110
Pro Lys Glu Tyr Thr Ala Gly Arg Glu Ala Asp Asp Ile Val Asn Trp
115 120 125
Leu Lys Lys Arg Thr Gly Pro Ala Ala Thr Thr Leu Pro Asp Gly Ala
130 135 140
Ala Ala Glu Ser Leu Val Glu Ser Ser Glu Val Ala Val Ile Gly Phe
145 150 155 160
Phe Lys Asp Val Glu Ser Asp Ser Ala Lys Gln Phe Leu Gln Ala Ala
165 170 175
Glu Ala Ile Asp Asp Ile Pro Phe Gly Ile Thr Ser Asn Ser Asp Val
180 185 190
Phe Ser Lys Tyr Gln Leu Asp Lys Asp Gly Val Val Leu Phe Lys Lys
195 200 205
Phe Asp Glu Gly Arg Asn Asn Phe Glu Gly Glu Val Thr Lys Glu Asn
210 215 220
Leu Leu Asp Phe Ile Lys His Asn Gln Leu Pro Leu Val Ile Glu Phe
225 230 235 240
Thr Glu Gln Thr Ala Pro Lys Ile Phe Gly Gly Glu Ile Lys Thr His
245 250 255
Ile Leu Leu Phe Leu Pro Lys Ser Val Ser Asp Tyr Asp Gly Lys Leu
260 265 270
Ser Asn Phe Lys Thr Ala Ala Glu Ser Phe Lys Gly Lys Ile Leu Phe
275 280 285
Ile Phe Ile Asp Ser Asp His Thr Asp Asn Gln Arg Ile Leu Glu Phe
290 295 300
Phe Gly Leu Lys Lys Glu Glu Cys Pro Ala Val Arg Leu Ile Thr Leu
305 310 315 320
Glu Glu Glu Met Thr Lys Tyr Lys Pro Glu Ser Glu Glu Leu Thr Ala
325 330 335
Glu Arg Ile Thr Glu Phe Cys His Arg Phe Leu Glu Gly Lys Ile Lys
340 345 350
Pro His Leu Met Ser Gln Glu Leu Pro Glu Asp Trp Asp Lys Gln Pro
355 360 365
Val Lys Val Leu Val Gly Lys Asn Phe Glu Asp Val Ala Phe Asp Glu
370 375 380
Lys Lys Asn Val Phe Val Glu Phe Tyr Ala Pro Trp Cys Gly His Cys
385 390 395 400
Lys Gln Leu Ala Pro Ile Trp Asp Lys Leu Gly Glu Thr Tyr Lys Asp
405 410 415
His Glu Asn Ile Val Ile Ala Lys Met Asp Ser Thr Ala Asn Glu Val
420 425 430
Glu Ala Val Lys Val His Ser Phe Pro Thr Leu Lys Phe Phe Pro Ala
435 440 445
Ser Ala Asp Arg Thr Val Ile Asp Tyr Asn Gly Glu Arg Thr Leu Asp
450 455 460
Gly Phe Lys Lys Phe Leu Glu Ser Gly Gly Gln Asp Gly Ala Gly Asp
465 470 475 480
Asp Asp Asp Leu Glu Asp Leu Glu Glu Ala Glu Glu Pro Asp Met Glu
485 490 495
Glu Asp Asp Asp Gln Lys Ala Val Lys Asp Glu Leu
500 505
<210> 44
<211> 603
<212> ДНК
<213> Homo sapiens
<220>
<221> misc_feature
<223> hGILZ
<400> 44
atggcccagt ccaagctcga ttgccgctca cctgtcggcc tcgactgctg caactgctgc 60
ctggacctgg cccatcggag tgggctccag cgaggcagca gcggggagaa caacaacccg 120
ggcagcccta cagtgagcaa ctttcggcag ctgcaggaaa agctggtctt tgagaacctc 180
aataccgaca agctcaacag cataatgcgg caggattcgc tagagccggt gctgcgggac 240
ccctgctacc tgatcaacga gggcatctgc aaccgcaaca tcgaccagac catgctctcc 300
atcctgctct tcttccacag tgcctccgga gccagcgtgg tggccataga caacaagatc 360
gaacaggcca tggatctggt gaagaatcat ctgatgtatg ctgtgagaga ggaggtggag 420
atcctgaagg agcagatccg agagctggtg gagaagaact cccagctaga gcgtgagaac 480
accctgttga agaccctggc aagcccagag cagctggaga agttccagtc ctgtctgagc 540
cctgaagagc cagctcccga atccccacaa gtgcccgagg cccctggtgg ttctgcggtg 600
taa 603
<210> 45
<211> 200
<212> ПРТ
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<223> hGILZ
<400> 45
Met Ala Gln Ser Lys Leu Asp Cys Arg Ser Pro Val Gly Leu Asp Cys
1 5 10 15
Cys Asn Cys Cys Leu Asp Leu Ala His Arg Ser Gly Leu Gln Arg Gly
20 25 30
Ser Ser Gly Glu Asn Asn Asn Pro Gly Ser Pro Thr Val Ser Asn Phe
35 40 45
Arg Gln Leu Gln Glu Lys Leu Val Phe Glu Asn Leu Asn Thr Asp Lys
50 55 60
Leu Asn Ser Ile Met Arg Gln Asp Ser Leu Glu Pro Val Leu Arg Asp
65 70 75 80
Pro Cys Tyr Leu Ile Asn Glu Gly Ile Cys Asn Arg Asn Ile Asp Gln
85 90 95
Thr Met Leu Ser Ile Leu Leu Phe Phe His Ser Ala Ser Gly Ala Ser
100 105 110
Val Val Ala Ile Asp Asn Lys Ile Glu Gln Ala Met Asp Leu Val Lys
115 120 125
Asn His Leu Met Tyr Ala Val Arg Glu Glu Val Glu Ile Leu Lys Glu
130 135 140
Gln Ile Arg Glu Leu Val Glu Lys Asn Ser Gln Leu Glu Arg Glu Asn
145 150 155 160
Thr Leu Leu Lys Thr Leu Ala Ser Pro Glu Gln Leu Glu Lys Phe Gln
165 170 175
Ser Cys Leu Ser Pro Glu Glu Pro Ala Pro Glu Ser Pro Gln Val Pro
180 185 190
Glu Ala Pro Gly Gly Ser Ala Val
195 200
<210> 46
<211> 651
<212> ДНК
<213> Homo sapiens
<220>
<221> misc_feature
<223> hCyPB
<400> 46
atgctgcgcc tctccgaacg caacatgaag gtgctccttg ccgccgccct catcgcgggg 60
tccgtcttct tcctgctgct gccgggacct tctgcggccg atgagaagaa gaaggggccc 120
aaagtcaccg tcaaggtgta ttttgaccta cgaattggag atgaagatgt aggccgggtg 180
atctttggtc tcttcggaaa gactgttcca aaaacagtgg ataattttgt ggccttagct 240
acaggagaga aaggatttgg ctacaaaaac agcaaattcc atcgtgtaat caaggacttc 300
atgatccagg gcggagactt caccagggga gatggcacag gaggaaagag catctacggt 360
gagcgcttcc ccgatgagaa cttcaaactg aagcactacg ggcctggctg ggtgagcatg 420
gccaacgcag gcaaagacac caacggctcc cagttcttca tcacgacagt caagacagcc 480
tggctagatg gcaagcatgt ggtgtttggc aaagttctag agggcatgga ggtggtgcgg 540
aaggtggaga gcaccaagac agacagccgg gataaacccc tgaaggatgt gatcatcgca 600
gactgcggca agatcgaggt ggagaagccc tttgccatcg ccaaggagta g 651
<210> 47
<211> 216
<212> ПРТ
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<223> hCyPB
<400> 47
Met Leu Arg Leu Ser Glu Arg Asn Met Lys Val Leu Leu Ala Ala Ala
1 5 10 15
Leu Ile Ala Gly Ser Val Phe Phe Leu Leu Leu Pro Gly Pro Ser Ala
20 25 30
Ala Asp Glu Lys Lys Lys Gly Pro Lys Val Thr Val Lys Val Tyr Phe
35 40 45
Asp Leu Arg Ile Gly Asp Glu Asp Val Gly Arg Val Ile Phe Gly Leu
50 55 60
Phe Gly Lys Thr Val Pro Lys Thr Val Asp Asn Phe Val Ala Leu Ala
65 70 75 80
Thr Gly Glu Lys Gly Phe Gly Tyr Lys Asn Ser Lys Phe His Arg Val
85 90 95
Ile Lys Asp Phe Met Ile Gln Gly Gly Asp Phe Thr Arg Gly Asp Gly
100 105 110
Thr Gly Gly Lys Ser Ile Tyr Gly Glu Arg Phe Pro Asp Glu Asn Phe
115 120 125
Lys Leu Lys His Tyr Gly Pro Gly Trp Val Ser Met Ala Asn Ala Gly
130 135 140
Lys Asp Thr Asn Gly Ser Gln Phe Phe Ile Thr Thr Val Lys Thr Ala
145 150 155 160
Trp Leu Asp Gly Lys His Val Val Phe Gly Lys Val Leu Glu Gly Met
165 170 175
Glu Val Val Arg Lys Val Glu Ser Thr Lys Thr Asp Ser Arg Asp Lys
180 185 190
Pro Leu Lys Asp Val Ile Ile Ala Asp Cys Gly Lys Ile Glu Val Glu
195 200 205
Lys Pro Phe Ala Ile Ala Lys Glu
210 215
<210> 48
<211> 1818
<212> ДНК
<213> Homo sapiens
<220>
<221> misc_feature
<223> hNRF2
<400> 48
atgatggact tggagctgcc gccgccggga ctcccgtccc agcaggacat ggatttgatt 60
gacatacttt ggaggcaaga tatagatctt ggagtaagtc gagaagtatt tgacttcagt 120
cagcgacgga aagagtatga gctggaaaaa cagaaaaaac ttgaaaagga aagacaagaa 180
caactccaaa aggagcaaga gaaagccttt ttcgctcagt tacaactaga tgaagagaca 240
ggtgaatttc tcccaattca gccagcccag cacatccagt cagaaaccag tggatctgcc 300
aactactccc aggttgccca cattcccaaa tcagatgctt tgtactttga tgactgcatg 360
cagcttttgg cgcagacatt cccgtttgta gatgacaatg aggtttcttc ggctacgttt 420
cagtcacttg ttcctgatat tcccggtcac atcgagagcc cagtcttcat tgctactaat 480
caggctcagt cacctgaaac ttctgttgct caggtagccc ctgttgattt agacggtatg 540
caacaggaca ttgagcaagt ttgggaggag ctattatcca ttcctgagtt acagtgtctt 600
aatattgaaa atgacaagct ggttgagact accatggttc caagtccaga agccaaactg 660
acagaagttg acaattatca tttttactca tctataccct caatggaaaa agaagtaggt 720
aactgtagtc cacattttct taatgctttt gaggattcct tcagcagcat cctctccaca 780
gaagacccca accagttgac agtgaactca ttaaattcag atgccacagt caacacagat 840
tttggtgatg aattttattc tgctttcata gctgagccca gtatcagcaa cagcatgccc 900
tcacctgcta ctttaagcca ttcactctct gaacttctaa atgggcccat tgatgtttct 960
gatctatcac tttgcaaagc tttcaaccaa aaccaccctg aaagcacagc agaattcaat 1020
gattctgact ccggcatttc actaaacaca agtcccagtg tggcatcacc agaacactca 1080
gtggaatctt ccagctatgg agacacacta cttggcctca gtgattctga agtggaagag 1140
ctagatagtg cccctggaag tgtcaaacag aatggtccta aaacaccagt acattcttct 1200
ggggatatgg tacaaccctt gtcaccatct caggggcaga gcactcacgt gcatgatgcc 1260
caatgtgaga acacaccaga gaaagaattg cctgtaagtc ctggtcatcg gaaaacccca 1320
ttcacaaaag acaaacattc aagccgcttg gaggctcatc tcacaagaga tgaacttagg 1380
gcaaaagctc tccatatccc attccctgta gaaaaaatca ttaacctccc tgttgttgac 1440
ttcaacgaaa tgatgtccaa agagcagttc aatgaagctc aacttgcatt aattcgggat 1500
atacgtagga ggggtaagaa taaagtggct gctcagaatt gcagaaaaag aaaactggaa 1560
aatatagtag aactagagca agatttagat catttgaaag atgaaaaaga aaaattgctc 1620
aaagaaaaag gagaaaatga caaaagcctt cacctactga aaaaacaact cagcacctta 1680
tatctcgaag ttttcagcat gctacgtgat gaagatggaa aaccttattc tcctagtgaa 1740
tactccctgc agcaaacaag agatggcaat gttttccttg ttcccaaaag taagaagcca 1800
gatgttaaga aaaactag 1818
<210> 49
<211> 605
<212> ПРТ
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<223> hNRF2
<400> 49
Met Met Asp Leu Glu Leu Pro Pro Pro Gly Leu Pro Ser Gln Gln Asp
1 5 10 15
Met Asp Leu Ile Asp Ile Leu Trp Arg Gln Asp Ile Asp Leu Gly Val
20 25 30
Ser Arg Glu Val Phe Asp Phe Ser Gln Arg Arg Lys Glu Tyr Glu Leu
35 40 45
Glu Lys Gln Lys Lys Leu Glu Lys Glu Arg Gln Glu Gln Leu Gln Lys
50 55 60
Glu Gln Glu Lys Ala Phe Phe Ala Gln Leu Gln Leu Asp Glu Glu Thr
65 70 75 80
Gly Glu Phe Leu Pro Ile Gln Pro Ala Gln His Ile Gln Ser Glu Thr
85 90 95
Ser Gly Ser Ala Asn Tyr Ser Gln Val Ala His Ile Pro Lys Ser Asp
100 105 110
Ala Leu Tyr Phe Asp Asp Cys Met Gln Leu Leu Ala Gln Thr Phe Pro
115 120 125
Phe Val Asp Asp Asn Glu Val Ser Ser Ala Thr Phe Gln Ser Leu Val
130 135 140
Pro Asp Ile Pro Gly His Ile Glu Ser Pro Val Phe Ile Ala Thr Asn
145 150 155 160
Gln Ala Gln Ser Pro Glu Thr Ser Val Ala Gln Val Ala Pro Val Asp
165 170 175
Leu Asp Gly Met Gln Gln Asp Ile Glu Gln Val Trp Glu Glu Leu Leu
180 185 190
Ser Ile Pro Glu Leu Gln Cys Leu Asn Ile Glu Asn Asp Lys Leu Val
195 200 205
Glu Thr Thr Met Val Pro Ser Pro Glu Ala Lys Leu Thr Glu Val Asp
210 215 220
Asn Tyr His Phe Tyr Ser Ser Ile Pro Ser Met Glu Lys Glu Val Gly
225 230 235 240
Asn Cys Ser Pro His Phe Leu Asn Ala Phe Glu Asp Ser Phe Ser Ser
245 250 255
Ile Leu Ser Thr Glu Asp Pro Asn Gln Leu Thr Val Asn Ser Leu Asn
260 265 270
Ser Asp Ala Thr Val Asn Thr Asp Phe Gly Asp Glu Phe Tyr Ser Ala
275 280 285
Phe Ile Ala Glu Pro Ser Ile Ser Asn Ser Met Pro Ser Pro Ala Thr
290 295 300
Leu Ser His Ser Leu Ser Glu Leu Leu Asn Gly Pro Ile Asp Val Ser
305 310 315 320
Asp Leu Ser Leu Cys Lys Ala Phe Asn Gln Asn His Pro Glu Ser Thr
325 330 335
Ala Glu Phe Asn Asp Ser Asp Ser Gly Ile Ser Leu Asn Thr Ser Pro
340 345 350
Ser Val Ala Ser Pro Glu His Ser Val Glu Ser Ser Ser Tyr Gly Asp
355 360 365
Thr Leu Leu Gly Leu Ser Asp Ser Glu Val Glu Glu Leu Asp Ser Ala
370 375 380
Pro Gly Ser Val Lys Gln Asn Gly Pro Lys Thr Pro Val His Ser Ser
385 390 395 400
Gly Asp Met Val Gln Pro Leu Ser Pro Ser Gln Gly Gln Ser Thr His
405 410 415
Val His Asp Ala Gln Cys Glu Asn Thr Pro Glu Lys Glu Leu Pro Val
420 425 430
Ser Pro Gly His Arg Lys Thr Pro Phe Thr Lys Asp Lys His Ser Ser
435 440 445
Arg Leu Glu Ala His Leu Thr Arg Asp Glu Leu Arg Ala Lys Ala Leu
450 455 460
His Ile Pro Phe Pro Val Glu Lys Ile Ile Asn Leu Pro Val Val Asp
465 470 475 480
Phe Asn Glu Met Met Ser Lys Glu Gln Phe Asn Glu Ala Gln Leu Ala
485 490 495
Leu Ile Arg Asp Ile Arg Arg Arg Gly Lys Asn Lys Val Ala Ala Gln
500 505 510
Asn Cys Arg Lys Arg Lys Leu Glu Asn Ile Val Glu Leu Glu Gln Asp
515 520 525
Leu Asp His Leu Lys Asp Glu Lys Glu Lys Leu Leu Lys Glu Lys Gly
530 535 540
Glu Asn Asp Lys Ser Leu His Leu Leu Lys Lys Gln Leu Ser Thr Leu
545 550 555 560
Tyr Leu Glu Val Phe Ser Met Leu Arg Asp Glu Asp Gly Lys Pro Tyr
565 570 575
Ser Pro Ser Glu Tyr Ser Leu Gln Gln Thr Arg Asp Gly Asn Val Phe
580 585 590
Leu Val Pro Lys Ser Lys Lys Pro Asp Val Lys Lys Asn
595 600 605
<210> 50
<211> 2754
<212> ДНК
<213> Homo sapiens
<220>
<221> misc_feature
<223> hHK1
<400> 50
atgatcgccg cgcagctcct ggcctattac ttcacggagc tgaaggatga ccaggtcaaa 60
aagattgaca agtatctcta tgccatgcgg ctctccgatg aaactctcat agatatcatg 120
actcgcttca ggaaggagat gaagaatggc ctctcccggg attttaatcc aacagccaca 180
gtcaagatgt tgccaacatt cgtaaggtcc attcctgatg gctctgaaaa gggagatttc 240
attgccctgg atcttggtgg gtcttccttt cgaattctgc gggtgcaagt gaatcatgag 300
aaaaaccaga atgttcacat ggagtccgag gtttatgaca ccccagagaa catcgtgcac 360
ggcagtggaa gccagctttt tgatcatgtt gctgagtgcc tgggagattt catggagaaa 420
aggaagatca aggacaagaa gttacctgtg ggattcacgt tttcttttcc ttgccaacaa 480
tccaaaatag atgaggccat cctgatcacc tggacaaagc gatttaaagc gagcggagtg 540
gaaggagcag atgtggtcaa actgcttaac aaagccatca aaaagcgagg ggactatgat 600
gccaacatcg tagctgtggt gaatgacaca gtgggcacca tgatgacctg tggctatgac 660
gaccagcact gtgaagtcgg cctgatcatc ggcactggca ccaatgcttg ctacatggag 720
gaactgaggc acattgatct ggtggaagga gacgagggga ggatgtgtat caatacagaa 780
tggggagcct ttggagacga tggatcatta gaagacatcc ggacagagtt tgacagggag 840
atagaccggg gatccctcaa ccctggaaaa cagctgtttg agaagatggt cagtggcatg 900
tacttgggag agctggttcg actgatccta gtcaagatgg ccaaggaggg cctcttattt 960
gaagggcgga tcaccccgga gctgctcacc cgagggaagt ttaacaccag tgatgtgtca 1020
gccatcgaaa agaataagga aggcctccac aatgccaaag aaatcctgac ccgcctggga 1080
gtggagccgt ccgatgatga ctgtgtctca gtccagcacg tttgcaccat tgtctcattt 1140
cgctcagcca acttggtggc tgccacactg ggcgccatct tgaaccgcct gcgtgataac 1200
aagggcacac ccaggctgcg gaccacggtt ggtgtcgacg gatctcttta caagacgcac 1260
ccacagtatt cccggcgttt ccacaagact ctaaggcgct tggtgccaga ctccgatgtg 1320
cgcttcctcc tctcggagag tggcagcggc aagggggctg ccatggtgac ggcggtggcc 1380
taccgcttgg ccgagcagca ccggcagata gaggagaccc tggctcattt ccacctcacc 1440
aaggacatgc tgctggaggt gaagaagagg atgcgggccg agatggagct ggggctgagg 1500
aagcagacgc acaacaatgc cgtggttaag atgctgccct ccttcgtccg gagaactccc 1560
gacgggaccg agaatggtga cttcttggcc ctggatcttg gaggaaccaa tttccgtgtg 1620
ctgctggtga aaatccgtag tgggaaaaag agaacggtgg aaatgcacaa caagatctac 1680
gccattccta ttgaaatcat gcagggcact ggggaagagc tgtttgatca cattgtctcc 1740
tgcatctctg acttcttgga ctacatgggg atcaaaggcc ccaggatgcc tctgggcttc 1800
acgttctcat ttccctgcca gcagacgagt ctggacgcgg gaatcttgat cacgtggaca 1860
aagggtttta aggcaacaga ctgcgtgggc cacgatgtag tcaccttact aagggatgcg 1920
ataaaaagga gagaggaatt tgacctggac gtggtggctg tggtcaacga cacagtgggc 1980
accatgatga cctgtgctta tgaggagccc acctgtgagg ttggactcat tgttgggacc 2040
ggcagcaatg cctgctacat ggaggagatg aagaacgtgg agatggtgga gggggaccag 2100
gggcagatgt gcatcaacat ggagtggggg gcctttgggg acaacgggtg tctggatgat 2160
atcaggacac actacgacag actggtggac gaatattccc taaatgctgg gaaacaaagg 2220
tatgagaaga tgatcagtgg tatgtacctg ggtgaaatcg tccgcaacat cttaatcgac 2280
ttcaccaaga agggattcct cttccgaggg cagatctctg agacgctgaa gacccggggc 2340
atctttgaga ccaagtttct ctctcagatc gagagtgacc gattagcact gctccaggtc 2400
cgggctatcc tccagcagct aggtctgaat agcacctgcg atgacagtat cctcgtcaag 2460
acagtgtgcg gggtggtgtc caggagggcc gcacagctgt gtggcgcagg catggctgcg 2520
gttgtggata agatccgcga gaacagagga ctggaccgtc tgaatgtgac tgtgggagtg 2580
gacgggacac tctacaagct tcatccacac ttctccagaa tcatgcacca gacggtgaag 2640
gaactgtcac caaaatgtaa cgtgtccttc ctcctgtctg aggatggcag cggcaagggg 2700
gccgccctca tcacggccgt gggcgtgcgg ttacgcacag aggcaagcag ctaa 2754
<210> 51
<211> 917
<212> ПРТ
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<223> hHK1
<400> 51
Met Ile Ala Ala Gln Leu Leu Ala Tyr Tyr Phe Thr Glu Leu Lys Asp
1 5 10 15
Asp Gln Val Lys Lys Ile Asp Lys Tyr Leu Tyr Ala Met Arg Leu Ser
20 25 30
Asp Glu Thr Leu Ile Asp Ile Met Thr Arg Phe Arg Lys Glu Met Lys
35 40 45
Asn Gly Leu Ser Arg Asp Phe Asn Pro Thr Ala Thr Val Lys Met Leu
50 55 60
Pro Thr Phe Val Arg Ser Ile Pro Asp Gly Ser Glu Lys Gly Asp Phe
65 70 75 80
Ile Ala Leu Asp Leu Gly Gly Ser Ser Phe Arg Ile Leu Arg Val Gln
85 90 95
Val Asn His Glu Lys Asn Gln Asn Val His Met Glu Ser Glu Val Tyr
100 105 110
Asp Thr Pro Glu Asn Ile Val His Gly Ser Gly Ser Gln Leu Phe Asp
115 120 125
His Val Ala Glu Cys Leu Gly Asp Phe Met Glu Lys Arg Lys Ile Lys
130 135 140
Asp Lys Lys Leu Pro Val Gly Phe Thr Phe Ser Phe Pro Cys Gln Gln
145 150 155 160
Ser Lys Ile Asp Glu Ala Ile Leu Ile Thr Trp Thr Lys Arg Phe Lys
165 170 175
Ala Ser Gly Val Glu Gly Ala Asp Val Val Lys Leu Leu Asn Lys Ala
180 185 190
Ile Lys Lys Arg Gly Asp Tyr Asp Ala Asn Ile Val Ala Val Val Asn
195 200 205
Asp Thr Val Gly Thr Met Met Thr Cys Gly Tyr Asp Asp Gln His Cys
210 215 220
Glu Val Gly Leu Ile Ile Gly Thr Gly Thr Asn Ala Cys Tyr Met Glu
225 230 235 240
Glu Leu Arg His Ile Asp Leu Val Glu Gly Asp Glu Gly Arg Met Cys
245 250 255
Ile Asn Thr Glu Trp Gly Ala Phe Gly Asp Asp Gly Ser Leu Glu Asp
260 265 270
Ile Arg Thr Glu Phe Asp Arg Glu Ile Asp Arg Gly Ser Leu Asn Pro
275 280 285
Gly Lys Gln Leu Phe Glu Lys Met Val Ser Gly Met Tyr Leu Gly Glu
290 295 300
Leu Val Arg Leu Ile Leu Val Lys Met Ala Lys Glu Gly Leu Leu Phe
305 310 315 320
Glu Gly Arg Ile Thr Pro Glu Leu Leu Thr Arg Gly Lys Phe Asn Thr
325 330 335
Ser Asp Val Ser Ala Ile Glu Lys Asn Lys Glu Gly Leu His Asn Ala
340 345 350
Lys Glu Ile Leu Thr Arg Leu Gly Val Glu Pro Ser Asp Asp Asp Cys
355 360 365
Val Ser Val Gln His Val Cys Thr Ile Val Ser Phe Arg Ser Ala Asn
370 375 380
Leu Val Ala Ala Thr Leu Gly Ala Ile Leu Asn Arg Leu Arg Asp Asn
385 390 395 400
Lys Gly Thr Pro Arg Leu Arg Thr Thr Val Gly Val Asp Gly Ser Leu
405 410 415
Tyr Lys Thr His Pro Gln Tyr Ser Arg Arg Phe His Lys Thr Leu Arg
420 425 430
Arg Leu Val Pro Asp Ser Asp Val Arg Phe Leu Leu Ser Glu Ser Gly
435 440 445
Ser Gly Lys Gly Ala Ala Met Val Thr Ala Val Ala Tyr Arg Leu Ala
450 455 460
Glu Gln His Arg Gln Ile Glu Glu Thr Leu Ala His Phe His Leu Thr
465 470 475 480
Lys Asp Met Leu Leu Glu Val Lys Lys Arg Met Arg Ala Glu Met Glu
485 490 495
Leu Gly Leu Arg Lys Gln Thr His Asn Asn Ala Val Val Lys Met Leu
500 505 510
Pro Ser Phe Val Arg Arg Thr Pro Asp Gly Thr Glu Asn Gly Asp Phe
515 520 525
Leu Ala Leu Asp Leu Gly Gly Thr Asn Phe Arg Val Leu Leu Val Lys
530 535 540
Ile Arg Ser Gly Lys Lys Arg Thr Val Glu Met His Asn Lys Ile Tyr
545 550 555 560
Ala Ile Pro Ile Glu Ile Met Gln Gly Thr Gly Glu Glu Leu Phe Asp
565 570 575
His Ile Val Ser Cys Ile Ser Asp Phe Leu Asp Tyr Met Gly Ile Lys
580 585 590
Gly Pro Arg Met Pro Leu Gly Phe Thr Phe Ser Phe Pro Cys Gln Gln
595 600 605
Thr Ser Leu Asp Ala Gly Ile Leu Ile Thr Trp Thr Lys Gly Phe Lys
610 615 620
Ala Thr Asp Cys Val Gly His Asp Val Val Thr Leu Leu Arg Asp Ala
625 630 635 640
Ile Lys Arg Arg Glu Glu Phe Asp Leu Asp Val Val Ala Val Val Asn
645 650 655
Asp Thr Val Gly Thr Met Met Thr Cys Ala Tyr Glu Glu Pro Thr Cys
660 665 670
Glu Val Gly Leu Ile Val Gly Thr Gly Ser Asn Ala Cys Tyr Met Glu
675 680 685
Glu Met Lys Asn Val Glu Met Val Glu Gly Asp Gln Gly Gln Met Cys
690 695 700
Ile Asn Met Glu Trp Gly Ala Phe Gly Asp Asn Gly Cys Leu Asp Asp
705 710 715 720
Ile Arg Thr His Tyr Asp Arg Leu Val Asp Glu Tyr Ser Leu Asn Ala
725 730 735
Gly Lys Gln Arg Tyr Glu Lys Met Ile Ser Gly Met Tyr Leu Gly Glu
740 745 750
Ile Val Arg Asn Ile Leu Ile Asp Phe Thr Lys Lys Gly Phe Leu Phe
755 760 765
Arg Gly Gln Ile Ser Glu Thr Leu Lys Thr Arg Gly Ile Phe Glu Thr
770 775 780
Lys Phe Leu Ser Gln Ile Glu Ser Asp Arg Leu Ala Leu Leu Gln Val
785 790 795 800
Arg Ala Ile Leu Gln Gln Leu Gly Leu Asn Ser Thr Cys Asp Asp Ser
805 810 815
Ile Leu Val Lys Thr Val Cys Gly Val Val Ser Arg Arg Ala Ala Gln
820 825 830
Leu Cys Gly Ala Gly Met Ala Ala Val Val Asp Lys Ile Arg Glu Asn
835 840 845
Arg Gly Leu Asp Arg Leu Asn Val Thr Val Gly Val Asp Gly Thr Leu
850 855 860
Tyr Lys Leu His Pro His Phe Ser Arg Ile Met His Gln Thr Val Lys
865 870 875 880
Glu Leu Ser Pro Lys Cys Asn Val Ser Phe Leu Leu Ser Glu Asp Gly
885 890 895
Ser Gly Lys Gly Ala Ala Leu Ile Thr Ala Val Gly Val Arg Leu Arg
900 905 910
Thr Glu Ala Ser Ser
915
<210> 52
<211> 1578
<212> ДНК
<213> Homo sapiens
<220>
<221> misc_feature
<223> hPDI
<400> 52
atgagccgcc agcttctgcc tgtactgctg ctgctgctgc tcagggcttc gtgcccatgg 60
ggtcaggaac agggagcgag gagcccctcg gaggagcctc cagaggagga aatccccaag 120
gaggatggga tcttggtgct gagccgccac accctgggcc tggccctgcg ggagcaccct 180
gccctgctgg tggaattcta tgccccgtgg tgtgggcact gccaggccct ggcccccgag 240
tacagcaagg cagctgccgt gctcgcggcc gagtcaatgg tggtcacgct ggccaaggtg 300
gatgggcccg cgcagcgcga gctggctgag gagtttggtg tgacggagta ccctacgctc 360
aagttcttcc gcaatgggaa ccgcacgcac ccggaggagt acacaggacc acgggacgct 420
gagggcattg ccgagtggct gcgacggcgg gtggggccca gtgccatgcg gctggaggac 480
gaggcggccg cccaggcgct gatcggtggc cgggacctag tggtcattgg cttcttccag 540
gacctgcagg acgaggacgt ggccaccttc ttggccttgg cccaggacgc cctggacatg 600
acctttggcc tcacagaccg gccgcggctc tttcagcagt ttggcctcac caaggacact 660
gtggttctct tcaagaagtt tgatgagggg cgggcagact tccccgtgga cgaggagctt 720
ggcctggacc tgggggatct gtcgcgcttc ctggtcacac acagcatgcg cctggtcacg 780
gagttcaaca gccagacgtc tgccaagatc ttcgcggcca ggatcctcaa ccacctgctg 840
ctgtttgtca accagacgct ggctgcgcac cgggagctcc tagcgggctt tggggaggca 900
gctccccgct tccgggggca ggtgctgttc gtggtggtgg acgtggcggc cgacaatgag 960
cacgtgctgc agtactttgg actcaaggct gaggcagccc ccactctgcg cttggtcaac 1020
cttgaaacca ctaagaagta tgcgcctgtg gatgggggcc ctgtcaccgc agcgtccatc 1080
actgctttct gccatgcagt cctcaacggc caagtcaagc cctatctcct gagccaggag 1140
ataccccctg attgggatca gcggccagtt aagaccctcg tgggcaagaa ttttgagcag 1200
gtggcttttg acgaaaccaa gaatgtgttt gtcaagttct atgccccgtg gtgcacccac 1260
tgcaaggaga tggcccctgc ctgggaggca ttggctgaga agtaccaaga ccacgaggac 1320
atcatcattg ctgagctgga tgccacggcc aacgagctgg atgccttcgc tgtgcacggc 1380
ttccctactc tcaagtactt cccagcaggg ccaggtcgga aggtgattga atacaaaagc 1440
accagggacc tggagacttt ctccaagttc ctggacaacg ggggcgtgct gcccacggag 1500
gagcccccgg aggagccagc agccccgttc ccggagccac cggccaactc cactatgggg 1560
tccaaggagg aactgtag 1578
<210> 53
<211> 525
<212> ПРТ
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<223> hPDI
<400> 53
Met Ser Arg Gln Leu Leu Pro Val Leu Leu Leu Leu Leu Leu Arg Ala
1 5 10 15
Ser Cys Pro Trp Gly Gln Glu Gln Gly Ala Arg Ser Pro Ser Glu Glu
20 25 30
Pro Pro Glu Glu Glu Ile Pro Lys Glu Asp Gly Ile Leu Val Leu Ser
35 40 45
Arg His Thr Leu Gly Leu Ala Leu Arg Glu His Pro Ala Leu Leu Val
50 55 60
Glu Phe Tyr Ala Pro Trp Cys Gly His Cys Gln Ala Leu Ala Pro Glu
65 70 75 80
Tyr Ser Lys Ala Ala Ala Val Leu Ala Ala Glu Ser Met Val Val Thr
85 90 95
Leu Ala Lys Val Asp Gly Pro Ala Gln Arg Glu Leu Ala Glu Glu Phe
100 105 110
Gly Val Thr Glu Tyr Pro Thr Leu Lys Phe Phe Arg Asn Gly Asn Arg
115 120 125
Thr His Pro Glu Glu Tyr Thr Gly Pro Arg Asp Ala Glu Gly Ile Ala
130 135 140
Glu Trp Leu Arg Arg Arg Val Gly Pro Ser Ala Met Arg Leu Glu Asp
145 150 155 160
Glu Ala Ala Ala Gln Ala Leu Ile Gly Gly Arg Asp Leu Val Val Ile
165 170 175
Gly Phe Phe Gln Asp Leu Gln Asp Glu Asp Val Ala Thr Phe Leu Ala
180 185 190
Leu Ala Gln Asp Ala Leu Asp Met Thr Phe Gly Leu Thr Asp Arg Pro
195 200 205
Arg Leu Phe Gln Gln Phe Gly Leu Thr Lys Asp Thr Val Val Leu Phe
210 215 220
Lys Lys Phe Asp Glu Gly Arg Ala Asp Phe Pro Val Asp Glu Glu Leu
225 230 235 240
Gly Leu Asp Leu Gly Asp Leu Ser Arg Phe Leu Val Thr His Ser Met
245 250 255
Arg Leu Val Thr Glu Phe Asn Ser Gln Thr Ser Ala Lys Ile Phe Ala
260 265 270
Ala Arg Ile Leu Asn His Leu Leu Leu Phe Val Asn Gln Thr Leu Ala
275 280 285
Ala His Arg Glu Leu Leu Ala Gly Phe Gly Glu Ala Ala Pro Arg Phe
290 295 300
Arg Gly Gln Val Leu Phe Val Val Val Asp Val Ala Ala Asp Asn Glu
305 310 315 320
His Val Leu Gln Tyr Phe Gly Leu Lys Ala Glu Ala Ala Pro Thr Leu
325 330 335
Arg Leu Val Asn Leu Glu Thr Thr Lys Lys Tyr Ala Pro Val Asp Gly
340 345 350
Gly Pro Val Thr Ala Ala Ser Ile Thr Ala Phe Cys His Ala Val Leu
355 360 365
Asn Gly Gln Val Lys Pro Tyr Leu Leu Ser Gln Glu Ile Pro Pro Asp
370 375 380
Trp Asp Gln Arg Pro Val Lys Thr Leu Val Gly Lys Asn Phe Glu Gln
385 390 395 400
Val Ala Phe Asp Glu Thr Lys Asn Val Phe Val Lys Phe Tyr Ala Pro
405 410 415
Trp Cys Thr His Cys Lys Glu Met Ala Pro Ala Trp Glu Ala Leu Ala
420 425 430
Glu Lys Tyr Gln Asp His Glu Asp Ile Ile Ile Ala Glu Leu Asp Ala
435 440 445
Thr Ala Asn Glu Leu Asp Ala Phe Ala Val His Gly Phe Pro Thr Leu
450 455 460
Lys Tyr Phe Pro Ala Gly Pro Gly Arg Lys Val Ile Glu Tyr Lys Ser
465 470 475 480
Thr Arg Asp Leu Glu Thr Phe Ser Lys Phe Leu Asp Asn Gly Gly Val
485 490 495
Leu Pro Thr Glu Glu Pro Pro Glu Glu Pro Ala Ala Pro Phe Pro Glu
500 505 510
Pro Pro Ala Asn Ser Thr Met Gly Ser Lys Glu Glu Leu
515 520 525
<210> 54
<211> 492
<212> ДНК
<213> Homo sapiens
<220>
<221> misc_feature
<223> hPIN1
<400> 54
atggcggacg aggagaagct gccgcccggc tgggagaagc gcatgagccg cagctcaggc 60
cgagtgtact acttcaacca catcactaac gccagccagt gggagcggcc cagcggcaac 120
agcagcagtg gtggcaaaaa cgggcagggg gagcctgcca gggtccgctg ctcgcacctg 180
ctggtgaagc acagccagtc acggcggccc tcgtcctggc ggcaggagaa gatcacccgg 240
accaaggagg aggccctgga gctgatcaac ggctacatcc agaagatcaa gtcgggagag 300
gaggactttg agtctctggc ctcacagttc agcgactgca gctcagccaa ggccagggga 360
gacctgggtg ccttcagcag aggtcagatg cagaagccat ttgaagacgc ctcgtttgcg 420
ctgcggacgg gggagatgag cgggcccgtg ttcacggatt ccggcatcca catcatcctc 480
cgcactgagt ga 492
<210> 55
<211> 163
<212> ПРТ
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<223> hPIN1
<400> 55
Met Ala Asp Glu Glu Lys Leu Pro Pro Gly Trp Glu Lys Arg Met Ser
1 5 10 15
Arg Ser Ser Gly Arg Val Tyr Tyr Phe Asn His Ile Thr Asn Ala Ser
20 25 30
Gln Trp Glu Arg Pro Ser Gly Asn Ser Ser Ser Gly Gly Lys Asn Gly
35 40 45
Gln Gly Glu Pro Ala Arg Val Arg Cys Ser His Leu Leu Val Lys His
50 55 60
Ser Gln Ser Arg Arg Pro Ser Ser Trp Arg Gln Glu Lys Ile Thr Arg
65 70 75 80
Thr Lys Glu Glu Ala Leu Glu Leu Ile Asn Gly Tyr Ile Gln Lys Ile
85 90 95
Lys Ser Gly Glu Glu Asp Phe Glu Ser Leu Ala Ser Gln Phe Ser Asp
100 105 110
Cys Ser Ser Ala Lys Ala Arg Gly Asp Leu Gly Ala Phe Ser Arg Gly
115 120 125
Gln Met Gln Lys Pro Phe Glu Asp Ala Ser Phe Ala Leu Arg Thr Gly
130 135 140
Glu Met Ser Gly Pro Val Phe Thr Asp Ser Gly Ile His Ile Ile Leu
145 150 155 160
Arg Thr Glu
<210> 56
<211> 264
<212> ДНК
<213> Homo sapiens
<220>
<221> misc_feature
<223> hSEPW1
<400> 56
atggctctcg ccgtccgagt cgtttattgt ggcgcttgag gctacaagtc caagtatctt 60
cagctcaaga agaagttaga agatgagttc cccggccgcc tggacatctg cggcgaggga 120
actccccagg ccaccgggtt ctttgaagtg atggtagccg ggaagttgat tcactctaag 180
aagaaaggcg atggctacgt ggacacagaa agcaagtttc tgaagttggt ggccgccatc 240
aaagccgcct tggctcaggg ctaa 264
<210> 57
<211> 87
<212> ПРТ
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<223> hSEPW1
<220>
<221> MISC_FEATURE
<222> (13)..(13)
<223> Особая аминокислота селеноцистеин (U)
<400> 57
Met Ala Leu Ala Val Arg Val Val Tyr Cys Gly Ala Xaa Gly Tyr Lys
1 5 10 15
Ser Lys Tyr Leu Gln Leu Lys Lys Lys Leu Glu Asp Glu Phe Pro Gly
20 25 30
Arg Leu Asp Ile Cys Gly Glu Gly Thr Pro Gln Ala Thr Gly Phe Phe
35 40 45
Glu Val Met Val Ala Gly Lys Leu Ile His Ser Lys Lys Lys Gly Asp
50 55 60
Gly Tyr Val Asp Thr Glu Ser Lys Phe Leu Lys Leu Val Ala Ala Ile
65 70 75 80
Lys Ala Ala Leu Ala Gln Gly
85
<210> 58
<211> 1254
<212> ДНК
<213> Homo sapiens
<220>
<221> misc_feature
<223> hCALR
<400> 58
atgctgctat ccgtgccgct gctgctcggc ctcctcggcc tggccgtcgc cgagcctgcc 60
gtctacttca aggagcagtt tctggacgga gacgggtgga cttcccgctg gatcgaatcc 120
aaacacaagt cagattttgg caaattcgtt ctcagttccg gcaagttcta cggtgacgag 180
gagaaagata aaggtttgca gacaagccag gatgcacgct tttatgctct gtcggccagt 240
ttcgagcctt tcagcaacaa aggccagacg ctggtggtgc agttcacggt gaaacatgag 300
cagaacatcg actgtggggg cggctatgtg aagctgtttc ctaatagttt ggaccagaca 360
gacatgcacg gagactcaga atacaacatc atgtttggtc ccgacatctg tggccctggc 420
accaagaagg ttcatgtcat cttcaactac aagggcaaga acgtgctgat caacaaggac 480
atccgttgca aggatgatga gtttacacac ctgtacacac tgattgtgcg gccagacaac 540
acctatgagg tgaagattga caacagccag gtggagtccg gctccttgga agacgattgg 600
gacttcctgc cacccaagaa gataaaggat cctgatgctt caaaaccgga agactgggat 660
gagcgggcca agatcgatga tcccacagac tccaagcctg aggactggga caagcccgag 720
catatccctg accctgatgc taagaagccc gaggactggg atgaagagat ggacggagag 780
tgggaacccc cagtgattca gaaccctgag tacaagggtg agtggaagcc ccggcagatc 840
gacaacccag attacaaggg cacttggatc cacccagaaa ttgacaaccc cgagtattct 900
cccgatccca gtatctatgc ctatgataac tttggcgtgc tgggcctgga cctctggcag 960
gtcaagtctg gcaccatctt tgacaacttc ctcatcacca acgatgaggc atacgctgag 1020
gagtttggca acgagacgtg gggcgtaaca aaggcagcag agaaacaaat gaaggacaaa 1080
caggacgagg agcagaggct taaggaggag gaagaagaca agaaacgcaa agaggaggag 1140
gaggcagagg acaaggagga tgatgaggac aaagatgagg atgaggagga tgaggaggac 1200
aaggaggaag atgaggagga agatgtcccc ggccaggcca aggacgagct gtag 1254
<210> 59
<211> 417
<212> ПРТ
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<223> hCALR
<400> 59
Met Leu Leu Ser Val Pro Leu Leu Leu Gly Leu Leu Gly Leu Ala Val
1 5 10 15
Ala Glu Pro Ala Val Tyr Phe Lys Glu Gln Phe Leu Asp Gly Asp Gly
20 25 30
Trp Thr Ser Arg Trp Ile Glu Ser Lys His Lys Ser Asp Phe Gly Lys
35 40 45
Phe Val Leu Ser Ser Gly Lys Phe Tyr Gly Asp Glu Glu Lys Asp Lys
50 55 60
Gly Leu Gln Thr Ser Gln Asp Ala Arg Phe Tyr Ala Leu Ser Ala Ser
65 70 75 80
Phe Glu Pro Phe Ser Asn Lys Gly Gln Thr Leu Val Val Gln Phe Thr
85 90 95
Val Lys His Glu Gln Asn Ile Asp Cys Gly Gly Gly Tyr Val Lys Leu
100 105 110
Phe Pro Asn Ser Leu Asp Gln Thr Asp Met His Gly Asp Ser Glu Tyr
115 120 125
Asn Ile Met Phe Gly Pro Asp Ile Cys Gly Pro Gly Thr Lys Lys Val
130 135 140
His Val Ile Phe Asn Tyr Lys Gly Lys Asn Val Leu Ile Asn Lys Asp
145 150 155 160
Ile Arg Cys Lys Asp Asp Glu Phe Thr His Leu Tyr Thr Leu Ile Val
165 170 175
Arg Pro Asp Asn Thr Tyr Glu Val Lys Ile Asp Asn Ser Gln Val Glu
180 185 190
Ser Gly Ser Leu Glu Asp Asp Trp Asp Phe Leu Pro Pro Lys Lys Ile
195 200 205
Lys Asp Pro Asp Ala Ser Lys Pro Glu Asp Trp Asp Glu Arg Ala Lys
210 215 220
Ile Asp Asp Pro Thr Asp Ser Lys Pro Glu Asp Trp Asp Lys Pro Glu
225 230 235 240
His Ile Pro Asp Pro Asp Ala Lys Lys Pro Glu Asp Trp Asp Glu Glu
245 250 255
Met Asp Gly Glu Trp Glu Pro Pro Val Ile Gln Asn Pro Glu Tyr Lys
260 265 270
Gly Glu Trp Lys Pro Arg Gln Ile Asp Asn Pro Asp Tyr Lys Gly Thr
275 280 285
Trp Ile His Pro Glu Ile Asp Asn Pro Glu Tyr Ser Pro Asp Pro Ser
290 295 300
Ile Tyr Ala Tyr Asp Asn Phe Gly Val Leu Gly Leu Asp Leu Trp Gln
305 310 315 320
Val Lys Ser Gly Thr Ile Phe Asp Asn Phe Leu Ile Thr Asn Asp Glu
325 330 335
Ala Tyr Ala Glu Glu Phe Gly Asn Glu Thr Trp Gly Val Thr Lys Ala
340 345 350
Ala Glu Lys Gln Met Lys Asp Lys Gln Asp Glu Glu Gln Arg Leu Lys
355 360 365
Glu Glu Glu Glu Asp Lys Lys Arg Lys Glu Glu Glu Glu Ala Glu Asp
370 375 380
Lys Glu Asp Asp Glu Asp Lys Asp Glu Asp Glu Glu Asp Glu Glu Asp
385 390 395 400
Lys Glu Glu Asp Glu Glu Glu Asp Val Pro Gly Gln Ala Lys Asp Glu
405 410 415
Leu
<210> 60
<211> 656
<212> ПРТ
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<223> DroBiP
<400> 60
Met Lys Phe Pro Met Val Ala Ala Ala Leu Leu Leu Leu Cys Ala Val
1 5 10 15
Arg Ala Glu Glu Lys Lys Glu Lys Asp Lys Glu Leu Gly Thr Val Ile
20 25 30
Gly Ile Asp Leu Gly Thr Thr Tyr Ser Cys Val Gly Val Tyr Lys Asn
35 40 45
Gly Arg Val Glu Ile Ile Ala Asn Asp Gln Gly Asn Arg Ile Thr Pro
50 55 60
Ser Tyr Val Ala Phe Thr Ala Asp Gly Glu Arg Leu Ile Gly Asp Ala
65 70 75 80
Ala Lys Asn Gln Leu Thr Thr Asn Pro Glu Asn Thr Val Phe Asp Ala
85 90 95
Lys Arg Leu Ile Gly Arg Glu Trp Ser Asp Thr Asn Val Gln His Asp
100 105 110
Ile Lys Phe Phe Pro Phe Lys Val Val Glu Lys Asn Ser Lys Pro His
115 120 125
Ile Ser Val Asp Thr Ser Gln Gly Ala Lys Val Phe Ala Pro Glu Glu
130 135 140
Ile Ser Ala Met Val Leu Gly Lys Met Lys Glu Thr Ala Glu Ala Tyr
145 150 155 160
Leu Gly Lys Lys Val Thr His Ala Val Val Thr Val Pro Ala Tyr Phe
165 170 175
Asn Asp Ala Gln Arg Gln Ala Thr Lys Asp Ala Gly Val Ile Ala Gly
180 185 190
Leu Gln Val Met Arg Ile Ile Asn Glu Pro Thr Ala Ala Ala Ile Ala
195 200 205
Tyr Gly Leu Asp Lys Lys Glu Gly Glu Lys Asn Val Leu Val Phe Asp
210 215 220
Leu Gly Gly Gly Thr Phe Asp Val Ser Leu Leu Thr Ile Asp Asn Gly
225 230 235 240
Val Phe Glu Val Val Ala Thr Asn Gly Asp Thr His Leu Gly Gly Glu
245 250 255
Asp Phe Asp Gln Arg Val Met Asp His Phe Ile Lys Leu Tyr Lys Lys
260 265 270
Lys Lys Gly Lys Asp Ile Arg Lys Asp Asn Arg Ala Val Gln Lys Leu
275 280 285
Arg Arg Glu Val Glu Lys Ala Lys Arg Ala Leu Ser Gly Ser His Gln
290 295 300
Val Arg Ile Glu Ile Glu Ser Phe Phe Glu Gly Asp Asp Phe Ser Glu
305 310 315 320
Thr Leu Thr Arg Ala Lys Phe Glu Glu Leu Asn Leu Asp Leu Phe Arg
325 330 335
Ser Thr Leu Lys Pro Val Gln Lys Val Leu Glu Asp Ala Asp Met Asn
340 345 350
Lys Lys Asp Val His Glu Ile Val Leu Val Gly Gly Ser Thr Arg Ile
355 360 365
Pro Lys Val Gln Gln Leu Val Lys Asp Phe Phe Gly Gly Lys Glu Pro
370 375 380
Ser Arg Gly Ile Asn Pro Asp Glu Ala Val Ala Tyr Gly Ala Ala Val
385 390 395 400
Gln Ala Gly Val Leu Ser Gly Glu Gln Asp Thr Asp Ala Ile Val Leu
405 410 415
Leu Asp Val Asn Pro Leu Thr Met Gly Ile Glu Thr Val Gly Gly Val
420 425 430
Met Thr Lys Leu Ile Pro Arg Asn Thr Val Ile Pro Thr Lys Lys Ser
435 440 445
Gln Val Phe Ser Thr Ala Ser Asp Asn Gln His Thr Val Thr Ile Gln
450 455 460
Val Tyr Glu Gly Glu Arg Pro Met Thr Lys Asp Asn His Leu Leu Gly
465 470 475 480
Lys Phe Asp Leu Thr Gly Ile Pro Pro Ala Pro Arg Gly Ile Pro Gln
485 490 495
Ile Glu Val Ser Phe Glu Ile Asp Ala Asn Gly Ile Leu Gln Val Ser
500 505 510
Ala Glu Asp Lys Gly Thr Gly Asn Lys Glu Lys Ile Val Ile Thr Asn
515 520 525
Asp Gln Asn Arg Leu Thr Pro Glu Asp Ile Asp Arg Met Ile Arg Asp
530 535 540
Ala Glu Lys Phe Ala Asp Glu Asp Lys Lys Leu Lys Glu Arg Val Glu
545 550 555 560
Ser Arg Asn Glu Leu Glu Ser Tyr Ala Tyr Ser Leu Lys Asn Gln Ile
565 570 575
Gly Asp Lys Asp Lys Leu Gly Ala Lys Leu Ser Asp Asp Glu Lys Asn
580 585 590
Lys Leu Glu Ser Ala Ile Asp Glu Ser Ile Lys Trp Leu Glu Gln Asn
595 600 605
Pro Asp Ala Asp Pro Glu Glu Tyr Lys Lys Gln Lys Lys Asp Leu Glu
610 615 620
Ala Ile Val Gln Pro Val Ile Ala Lys Leu Tyr Gln Gly Ala Gly Gly
625 630 635 640
Ala Pro Pro Pro Glu Gly Gly Asp Asp Ala Asp Leu Lys Asp Glu Leu
645 650 655
<210> 61
<211> 1371
<212> ДНК
<213> Homo sapiens
<220>
<221> misc_feature
<223> hDDOST
<400> 61
atggggtact tccggtgtgc aggtgctggg tccttcggca ggaggaggaa gatggagccc 60
agcaccgcgg cccgggcttg ggccctcttt tggttgctgc tgcccttgct tggcgcggtt 120
tgcgccagcg gaccccgcac cttagtgctg ctggacaacc tcaacgtgcg ggagactcat 180
tcgcttttct tccggagcct gaaggaccgg ggctttgagc tcacattcaa gaccgctgat 240
gaccccagcc tgtctctcat aaagtatggg gaattcctct atgacaatct catcattttc 300
tccccttcgg tagaagattt tggaggcaac atcaacgtgg agaccatcag tgcctttatt 360
gacggcggag gcagtgtgct ggtagctgcc agctccgaca ttggtgaccc tcttcgagag 420
ctgggcagtg agtgcgggat tgagtttgac gaggagaaaa cggctgtcat tgaccatcac 480
aactatgaca tctcagacct tggccagcat acgctcatcg tggctgacac tgagaacctg 540
ctgaaggccc caaccatcgt tgggaaatca tctctaaatc ccatcctctt tcgaggtgtt 600
gggatggtgg ccgatcctga taaccctttg gtgctggaca tcctgacggg ctcttccacc 660
tcttactcct tcttcccgga caagcctatc acccagtatc cacatgcggt ggggaagaac 720
accctcctca ttgctgggct ccaggccagg aacaatgccc gcgtcatctt cagcggctcc 780
ctcgacttct tcagcgactc cttcttcaac tcagcagtgc agaaggcggc gcccggctcc 840
cagaggtatt cccagacagg caactatgaa ctagctgtgg ccctctcccg ctgggtgttc 900
aaggaggagg gtgtcctccg tgtggggcct gtgtcccatc atcgggtggg cgagacagcc 960
ccacccaatg cctacactgt cactgaccta gtggagtata gcatcgtgat ccagcagctc 1020
tcaaatggca aatgggtccc ctttgatggc gatgacattc agctggagtt tgtccgcatt 1080
gatccttttg tgaggacctt cctgaagaag aaaggtggca aatacagtgt tcagttcaag 1140
ttgcccgacg tgtatggtgt attccagttt aaagtggatt acaaccggct aggctacaca 1200
cacctgtact cttccactca ggtatccgtg cggccactcc agcacacgca gtatgagcgc 1260
ttcatcccct cggcctaccc ctactacgcc agcgccttct ccatgatgct ggggctcttc 1320
atcttcagca tcgtcttctt gcacatgaag gagaaggaga agtccgactg a 1371
<210> 62
<211> 456
<212> ПРТ
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<223> hDDOST
<400> 62
Met Gly Tyr Phe Arg Cys Ala Gly Ala Gly Ser Phe Gly Arg Arg Arg
1 5 10 15
Lys Met Glu Pro Ser Thr Ala Ala Arg Ala Trp Ala Leu Phe Trp Leu
20 25 30
Leu Leu Pro Leu Leu Gly Ala Val Cys Ala Ser Gly Pro Arg Thr Leu
35 40 45
Val Leu Leu Asp Asn Leu Asn Val Arg Glu Thr His Ser Leu Phe Phe
50 55 60
Arg Ser Leu Lys Asp Arg Gly Phe Glu Leu Thr Phe Lys Thr Ala Asp
65 70 75 80
Asp Pro Ser Leu Ser Leu Ile Lys Tyr Gly Glu Phe Leu Tyr Asp Asn
85 90 95
Leu Ile Ile Phe Ser Pro Ser Val Glu Asp Phe Gly Gly Asn Ile Asn
100 105 110
Val Glu Thr Ile Ser Ala Phe Ile Asp Gly Gly Gly Ser Val Leu Val
115 120 125
Ala Ala Ser Ser Asp Ile Gly Asp Pro Leu Arg Glu Leu Gly Ser Glu
130 135 140
Cys Gly Ile Glu Phe Asp Glu Glu Lys Thr Ala Val Ile Asp His His
145 150 155 160
Asn Tyr Asp Ile Ser Asp Leu Gly Gln His Thr Leu Ile Val Ala Asp
165 170 175
Thr Glu Asn Leu Leu Lys Ala Pro Thr Ile Val Gly Lys Ser Ser Leu
180 185 190
Asn Pro Ile Leu Phe Arg Gly Val Gly Met Val Ala Asp Pro Asp Asn
195 200 205
Pro Leu Val Leu Asp Ile Leu Thr Gly Ser Ser Thr Ser Tyr Ser Phe
210 215 220
Phe Pro Asp Lys Pro Ile Thr Gln Tyr Pro His Ala Val Gly Lys Asn
225 230 235 240
Thr Leu Leu Ile Ala Gly Leu Gln Ala Arg Asn Asn Ala Arg Val Ile
245 250 255
Phe Ser Gly Ser Leu Asp Phe Phe Ser Asp Ser Phe Phe Asn Ser Ala
260 265 270
Val Gln Lys Ala Ala Pro Gly Ser Gln Arg Tyr Ser Gln Thr Gly Asn
275 280 285
Tyr Glu Leu Ala Val Ala Leu Ser Arg Trp Val Phe Lys Glu Glu Gly
290 295 300
Val Leu Arg Val Gly Pro Val Ser His His Arg Val Gly Glu Thr Ala
305 310 315 320
Pro Pro Asn Ala Tyr Thr Val Thr Asp Leu Val Glu Tyr Ser Ile Val
325 330 335
Ile Gln Gln Leu Ser Asn Gly Lys Trp Val Pro Phe Asp Gly Asp Asp
340 345 350
Ile Gln Leu Glu Phe Val Arg Ile Asp Pro Phe Val Arg Thr Phe Leu
355 360 365
Lys Lys Lys Gly Gly Lys Tyr Ser Val Gln Phe Lys Leu Pro Asp Val
370 375 380
Tyr Gly Val Phe Gln Phe Lys Val Asp Tyr Asn Arg Leu Gly Tyr Thr
385 390 395 400
His Leu Tyr Ser Ser Thr Gln Val Ser Val Arg Pro Leu Gln His Thr
405 410 415
Gln Tyr Glu Arg Phe Ile Pro Ser Ala Tyr Pro Tyr Tyr Ala Ser Ala
420 425 430
Phe Ser Met Met Leu Gly Leu Phe Ile Phe Ser Ile Val Phe Leu His
435 440 445
Met Lys Glu Lys Glu Lys Ser Asp
450 455
<210> 63
<211> 1023
<212> ДНК
<213> Homo sapiens
<220>
<221> misc_feature
<223> hHSP40
<400> 63
atgggtaaag actactacca gacgttgggc ctggcccgcg gcgcgtcgga cgaggagatc 60
aagcgggcct accgccgcca ggcgctgcgc taccacccgg acaagaacaa ggagcccggc 120
gccgaggaga agttcaagga gatcgctgag gcctacgacg tgctcagcga cccgcgcaag 180
cgcgagatct tcgaccgcta cggggaggaa ggcctaaagg ggagtggccc cagtggcggt 240
agcggcggtg gtgccaatgg tacctctttc agctacacat tccatggaga ccctcatgcc 300
atgtttgctg agttcttcgg tggcagaaat ccctttgaca ccttttttgg gcagcggaac 360
ggggaggaag gcatggacat tgatgaccca ttctctggct tccctatggg catgggtggc 420
ttcaccaacg tgaactttgg ccgctcccgc tctgcccaag agcccgcccg aaagaagcaa 480
gatcccccag tcacccacga ccttcgagtc tcccttgaag agatctacag cggctgtacc 540
aagaagatga aaatctccca caagcggcta aaccccgacg gaaagagcat tcgaaacgaa 600
gacaaaatat tgaccatcga agtgaagaag gggtggaaag aaggaaccaa aatcactttc 660
cccaaggaag gagaccagac ctccaacaac attccagctg atatcgtctt tgttttaaag 720
gacaagcccc acaatatctt taagagagat ggctctgatg tcatttatcc tgccaggatc 780
agcctccggg aggctctgtg tggctgcaca gtgaacgtcc ccactctgga cggcaggacg 840
atacccgtcg tattcaaaga tgttatcagg cctggcatgc ggcgaaaagt tcctggagaa 900
ggcctccccc tccccaaaac acccgagaaa cgtggggacc tcattattga gtttgaagtg 960
atcttccccg aaaggattcc ccagacatca agaaccgtac ttgagcaggt tcttccaata 1020
tag 1023
<210> 64
<211> 340
<212> ПРТ
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<223> hHSP40
<400> 64
Met Gly Lys Asp Tyr Tyr Gln Thr Leu Gly Leu Ala Arg Gly Ala Ser
1 5 10 15
Asp Glu Glu Ile Lys Arg Ala Tyr Arg Arg Gln Ala Leu Arg Tyr His
20 25 30
Pro Asp Lys Asn Lys Glu Pro Gly Ala Glu Glu Lys Phe Lys Glu Ile
35 40 45
Ala Glu Ala Tyr Asp Val Leu Ser Asp Pro Arg Lys Arg Glu Ile Phe
50 55 60
Asp Arg Tyr Gly Glu Glu Gly Leu Lys Gly Ser Gly Pro Ser Gly Gly
65 70 75 80
Ser Gly Gly Gly Ala Asn Gly Thr Ser Phe Ser Tyr Thr Phe His Gly
85 90 95
Asp Pro His Ala Met Phe Ala Glu Phe Phe Gly Gly Arg Asn Pro Phe
100 105 110
Asp Thr Phe Phe Gly Gln Arg Asn Gly Glu Glu Gly Met Asp Ile Asp
115 120 125
Asp Pro Phe Ser Gly Phe Pro Met Gly Met Gly Gly Phe Thr Asn Val
130 135 140
Asn Phe Gly Arg Ser Arg Ser Ala Gln Glu Pro Ala Arg Lys Lys Gln
145 150 155 160
Asp Pro Pro Val Thr His Asp Leu Arg Val Ser Leu Glu Glu Ile Tyr
165 170 175
Ser Gly Cys Thr Lys Lys Met Lys Ile Ser His Lys Arg Leu Asn Pro
180 185 190
Asp Gly Lys Ser Ile Arg Asn Glu Asp Lys Ile Leu Thr Ile Glu Val
195 200 205
Lys Lys Gly Trp Lys Glu Gly Thr Lys Ile Thr Phe Pro Lys Glu Gly
210 215 220
Asp Gln Thr Ser Asn Asn Ile Pro Ala Asp Ile Val Phe Val Leu Lys
225 230 235 240
Asp Lys Pro His Asn Ile Phe Lys Arg Asp Gly Ser Asp Val Ile Tyr
245 250 255
Pro Ala Arg Ile Ser Leu Arg Glu Ala Leu Cys Gly Cys Thr Val Asn
260 265 270
Val Pro Thr Leu Asp Gly Arg Thr Ile Pro Val Val Phe Lys Asp Val
275 280 285
Ile Arg Pro Gly Met Arg Arg Lys Val Pro Gly Glu Gly Leu Pro Leu
290 295 300
Pro Lys Thr Pro Glu Lys Arg Gly Asp Leu Ile Ile Glu Phe Glu Val
305 310 315 320
Ile Phe Pro Glu Arg Ile Pro Gln Thr Ser Arg Thr Val Leu Glu Gln
325 330 335
Val Leu Pro Ile
340
<210> 65
<211> 1662
<212> ДНК
<213> Homo sapiens
<220>
<221> misc_feature
<223> hATP5A1
<400> 65
atgctgtccg tgcgcgttgc tgcggccgtg gtccgcgccc ttcctcggcg ggccggactg 60
gtctccagaa atgctttggg ttcatctttc attgctgcaa ggaacttcca tgcctctaac 120
actcatcttc aaaagactgg gactgctgag atgtcctcta ttcttgaaga gcgtattctt 180
ggagctgata cctctgttga tcttgaagaa actgggcgtg tcttaagtat tggtgatggt 240
attgcccgcg tacatgggct gaggaatgtt caagcagaag aaatggtaga gttttcttca 300
ggcttaaagg gtatgtcctt gaacttggaa cctgacaatg ttggtgttgt cgtgtttgga 360
aatgataaac taattaagga aggagatata gtgaagagga caggagccat tgtggacgtt 420
ccagttggtg aggagctgtt gggtcgtgta gttgatgccc ttggtaatgc tattgatgga 480
aagggtccaa ttggttccaa gacgcgtagg cgagttggtc tgaaagcccc cggtatcatt 540
cctcgaattt cagtgcggga accaatgcag actggcatta aggctgtgga tagcttggtg 600
ccaattggtc gtggtcagcg tgaactgatt attggtgacc gacagactgg gaaaacctca 660
attgctattg acacaatcat taaccagaaa cgtttcaatg atggatctga tgaaaagaag 720
aagctgtact gtatttatgt tgctattggt caaaagagat ccactgttgc ccagttggtg 780
aagagactta cagatgcaga tgccatgaag tacaccattg tggtgtcggc tacggcctcg 840
gatgctgccc cacttcagta cctggctcct tactctggct gttccatggg agagtatttt 900
agagacaatg gcaaacatgc tttgatcatc tatgacgact tatccaaaca ggctgttgct 960
taccgtcaga tgtctctgtt gctccgccga ccccctggtc gtgaggccta tcctggtgat 1020
gtgttctacc tacactcccg gttgctggag agagcagcca aaatgaacga tgcttttggt 1080
ggtggctcct tgactgcttt gccagtcata gaaacacagg ctggtgatgt gtctgcttac 1140
attccaacaa atgtcatttc catcactgac ggacagatct tcttggaaac agaattgttc 1200
tacaaaggta tccgccctgc aattaacgtt ggtctgtctg tatctcgtgt cggatccgct 1260
gcccaaacca gggctatgaa gcaggtagca ggtaccatga agctggaatt ggctcagtat 1320
cgtgaggttg ctgcttttgc ccagttcggt tctgacctcg atgctgccac tcaacaactt 1380
ttgagtcgtg gcgtgcgtct aactgagttg ctgaagcaag gacagtattc tcccatggct 1440
attgaagaac aagtggctgt tatctatgcg ggtgtaaggg gatatcttga taaactggag 1500
cccagcaaga ttacaaagtt tgagaatgct ttcttgtctc atgtcgtcag ccagcaccaa 1560
gccttgttgg gcactatcag ggctgatgga aagatctcag aacaatcaga tgcaaagctg 1620
aaagagattg taacaaattt cttggctgga tttgaagctt aa 1662
<210> 66
<211> 553
<212> ПРТ
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<223> hATP5A1
<400> 66
Met Leu Ser Val Arg Val Ala Ala Ala Val Val Arg Ala Leu Pro Arg
1 5 10 15
Arg Ala Gly Leu Val Ser Arg Asn Ala Leu Gly Ser Ser Phe Ile Ala
20 25 30
Ala Arg Asn Phe His Ala Ser Asn Thr His Leu Gln Lys Thr Gly Thr
35 40 45
Ala Glu Met Ser Ser Ile Leu Glu Glu Arg Ile Leu Gly Ala Asp Thr
50 55 60
Ser Val Asp Leu Glu Glu Thr Gly Arg Val Leu Ser Ile Gly Asp Gly
65 70 75 80
Ile Ala Arg Val His Gly Leu Arg Asn Val Gln Ala Glu Glu Met Val
85 90 95
Glu Phe Ser Ser Gly Leu Lys Gly Met Ser Leu Asn Leu Glu Pro Asp
100 105 110
Asn Val Gly Val Val Val Phe Gly Asn Asp Lys Leu Ile Lys Glu Gly
115 120 125
Asp Ile Val Lys Arg Thr Gly Ala Ile Val Asp Val Pro Val Gly Glu
130 135 140
Glu Leu Leu Gly Arg Val Val Asp Ala Leu Gly Asn Ala Ile Asp Gly
145 150 155 160
Lys Gly Pro Ile Gly Ser Lys Thr Arg Arg Arg Val Gly Leu Lys Ala
165 170 175
Pro Gly Ile Ile Pro Arg Ile Ser Val Arg Glu Pro Met Gln Thr Gly
180 185 190
Ile Lys Ala Val Asp Ser Leu Val Pro Ile Gly Arg Gly Gln Arg Glu
195 200 205
Leu Ile Ile Gly Asp Arg Gln Thr Gly Lys Thr Ser Ile Ala Ile Asp
210 215 220
Thr Ile Ile Asn Gln Lys Arg Phe Asn Asp Gly Ser Asp Glu Lys Lys
225 230 235 240
Lys Leu Tyr Cys Ile Tyr Val Ala Ile Gly Gln Lys Arg Ser Thr Val
245 250 255
Ala Gln Leu Val Lys Arg Leu Thr Asp Ala Asp Ala Met Lys Tyr Thr
260 265 270
Ile Val Val Ser Ala Thr Ala Ser Asp Ala Ala Pro Leu Gln Tyr Leu
275 280 285
Ala Pro Tyr Ser Gly Cys Ser Met Gly Glu Tyr Phe Arg Asp Asn Gly
290 295 300
Lys His Ala Leu Ile Ile Tyr Asp Asp Leu Ser Lys Gln Ala Val Ala
305 310 315 320
Tyr Arg Gln Met Ser Leu Leu Leu Arg Arg Pro Pro Gly Arg Glu Ala
325 330 335
Tyr Pro Gly Asp Val Phe Tyr Leu His Ser Arg Leu Leu Glu Arg Ala
340 345 350
Ala Lys Met Asn Asp Ala Phe Gly Gly Gly Ser Leu Thr Ala Leu Pro
355 360 365
Val Ile Glu Thr Gln Ala Gly Asp Val Ser Ala Tyr Ile Pro Thr Asn
370 375 380
Val Ile Ser Ile Thr Asp Gly Gln Ile Phe Leu Glu Thr Glu Leu Phe
385 390 395 400
Tyr Lys Gly Ile Arg Pro Ala Ile Asn Val Gly Leu Ser Val Ser Arg
405 410 415
Val Gly Ser Ala Ala Gln Thr Arg Ala Met Lys Gln Val Ala Gly Thr
420 425 430
Met Lys Leu Glu Leu Ala Gln Tyr Arg Glu Val Ala Ala Phe Ala Gln
435 440 445
Phe Gly Ser Asp Leu Asp Ala Ala Thr Gln Gln Leu Leu Ser Arg Gly
450 455 460
Val Arg Leu Thr Glu Leu Leu Lys Gln Gly Gln Tyr Ser Pro Met Ala
465 470 475 480
Ile Glu Glu Gln Val Ala Val Ile Tyr Ala Gly Val Arg Gly Tyr Leu
485 490 495
Asp Lys Leu Glu Pro Ser Lys Ile Thr Lys Phe Glu Asn Ala Phe Leu
500 505 510
Ser His Val Val Ser Gln His Gln Ala Leu Leu Gly Thr Ile Arg Ala
515 520 525
Asp Gly Lys Ile Ser Glu Gln Ser Asp Ala Lys Leu Lys Glu Ile Val
530 535 540
Thr Asn Phe Leu Ala Gly Phe Glu Ala
545 550
<210> 67
<211> 3129
<212> ДНК
<213> Homo sapiens
<220>
<221> misc_feature
<223> hSERCA2
<400> 67
atggagaacg cgcacaccaa gacggtggag gaggtgctgg gccacttcgg cgtcaacgag 60
agtacggggc tgagcctgga acaggtcaag aagcttaagg agagatgggg ctccaacgag 120
ttaccggctg aagaaggaaa aaccttgctg gaacttgtga ttgagcagtt tgaagacttg 180
ctagttagga ttttattact ggcagcatgt atatcttttg ttttggcttg gtttgaagaa 240
ggtgaagaaa caattacagc ctttgtagaa ccttttgtaa ttttactcat attagtagcc 300
aatgcaattg tgggtgtatg gcaggaaaga aatgctgaaa atgccatcga agcccttaag 360
gaatatgagc ctgaaatggg caaagtgtat cgacaggaca gaaagagtgt gcagcggatt 420
aaagctaaag acatagttcc tggtgatatt gtagaaattg ctgttggtga caaagttcct 480
gctgatataa ggttaacttc catcaaatct accacactaa gagttgacca gtcaattctc 540
acaggtgaat ctgtctctgt catcaagcac actgatcccg tccctgaccc acgagctgtc 600
aaccaagata aaaagaacat gctgttttct ggtacaaaca ttgctgctgg gaaagctatg 660
ggagtggtgg tagcaactgg agttaacacc gaaattggca agatccggga tgaaatggtg 720
gcaacagaac aggagagaac accccttcag caaaaactag atgaatttgg ggaacagctt 780
tccaaagtca tctcccttat ttgcattgca gtctggatca taaatattgg gcacttcaat 840
gacccggttc atggagggtc ctggatcaga ggtgctattt actactttaa aattgcagtg 900
gccctggctg tagcagccat tcctgaaggt ctgcctgcag tcatcaccac ctgcctggct 960
cttggaactc gcagaatggc aaagaaaaat gccattgttc gaagcctccc gtctgtggaa 1020
acccttggtt gtacttctgt tatctgctca gacaagactg gtacacttac aacaaaccag 1080
atgtcagtct gcaggatgtt cattctggac agagtggaag gtgatacttg ttcccttaat 1140
gagtttacca taactggatc aacttatgca cctattggag aagtgcataa agatgataaa 1200
ccagtgaatt gtcaccagta tgatggtctg gtagaattag caacaatttg tgctctttgt 1260
aatgactctg ctttggatta caatgaggca aagggtgtgt atgaaaaagt tggagaagct 1320
acagagactg ctctcacttg cctagtagag aagatgaatg tatttgatac cgaattgaag 1380
ggtctttcta aaatagaacg tgcaaatgcc tgcaactcag tcattaaaca gctgatgaaa 1440
aaggaattca ctctagagtt ttcacgtgac agaaagtcaa tgtcggttta ctgtacacca 1500
aataaaccaa gcaggacatc aatgagcaag atgtttgtga agggtgctcc tgaaggtgtc 1560
attgacaggt gcacccacat tcgagttgga agtactaagg ttcctatgac ctctggagtc 1620
aaacagaaga tcatgtctgt cattcgagag tggggtagtg gcagcgacac actgcgatgc 1680
ctggccctgg ccactcatga caacccactg agaagagaag aaatgcacct tgaggactct 1740
gccaacttta ttaaatatga gaccaatctg accttcgttg gctgcgtggg catgctggat 1800
cctccgagaa tcgaggtggc ctcctccgtg aagctgtgcc ggcaagcagg catccgggtc 1860
atcatgatca ctggggacaa caagggcact gctgtggcca tctgtcgccg catcggcatc 1920
ttcgggcagg atgaggacgt gacgtcaaaa gctttcacag gccgggagtt tgatgaactc 1980
aacccctccg cccagcgaga cgcctgcctg aacgcccgct gttttgctcg agttgaaccc 2040
tcccacaagt ctaaaatcgt agaatttctt cagtcttttg atgagattac agctatgact 2100
ggcgatggcg tgaacgatgc tcctgctctg aagaaagccg agattggcat tgctatgggc 2160
tctggcactg cggtggctaa aaccgcctct gagatggtcc tggcggatga caacttctcc 2220
accattgtgg ctgccgttga ggaggggcgg gcaatctaca acaacatgaa acagttcatc 2280
cgctacctca tctcgtccaa cgtcggggaa gttgtctgta ttttcctgac agcagccctt 2340
ggatttcccg aggctttgat tcctgttcag ctgctctggg tcaatctggt gacagatggc 2400
ctgcctgcca ctgcactggg gttcaaccct cctgatctgg acatcatgaa taaacctccc 2460
cggaacccaa aggaaccatt gatcagcggg tggctctttt tccgttactt ggctattggc 2520
tgttacgtcg gcgctgctac cgtgggtgct gctgcatggt ggttcattgc tgctgacggt 2580
ggtccaagag tgtccttcta ccagctgagt catttcctac agtgtaaaga ggacaacccg 2640
gactttgaag gcgtggattg tgcaatcttt gaatccccat acccgatgac aatggcgctc 2700
tctgttctag taactataga aatgtgtaac gccctcaaca gcttgtccga aaaccagtcc 2760
ttgctgagga tgcccccctg ggagaacatc tggctcgtgg gctccatctg cctgtccatg 2820
tcactccact tcctgatcct ctatgtcgaa cccttgccac tcatcttcca gatcacaccg 2880
ctgaacgtga cccagtggct gatggtgctg aaaatctcct tgcccgtgat tctcatggat 2940
gagacgctca agtttgtggc ccgcaactac ctggaacctg gtaaagagtg tgtgcagcct 3000
gccaccaaat cctgctcgtt ctcggcatgc accgatggga tttcctggcc gtttgtgctg 3060
ctcataatgc ccctggtgat ctgggtctat agcacagaca ctaactttag cgatatgttc 3120
tggtcttga 3129
<210> 68
<211> 1042
<212> ПРТ
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<223> hSERCA2
<400> 68
Met Glu Asn Ala His Thr Lys Thr Val Glu Glu Val Leu Gly His Phe
1 5 10 15
Gly Val Asn Glu Ser Thr Gly Leu Ser Leu Glu Gln Val Lys Lys Leu
20 25 30
Lys Glu Arg Trp Gly Ser Asn Glu Leu Pro Ala Glu Glu Gly Lys Thr
35 40 45
Leu Leu Glu Leu Val Ile Glu Gln Phe Glu Asp Leu Leu Val Arg Ile
50 55 60
Leu Leu Leu Ala Ala Cys Ile Ser Phe Val Leu Ala Trp Phe Glu Glu
65 70 75 80
Gly Glu Glu Thr Ile Thr Ala Phe Val Glu Pro Phe Val Ile Leu Leu
85 90 95
Ile Leu Val Ala Asn Ala Ile Val Gly Val Trp Gln Glu Arg Asn Ala
100 105 110
Glu Asn Ala Ile Glu Ala Leu Lys Glu Tyr Glu Pro Glu Met Gly Lys
115 120 125
Val Tyr Arg Gln Asp Arg Lys Ser Val Gln Arg Ile Lys Ala Lys Asp
130 135 140
Ile Val Pro Gly Asp Ile Val Glu Ile Ala Val Gly Asp Lys Val Pro
145 150 155 160
Ala Asp Ile Arg Leu Thr Ser Ile Lys Ser Thr Thr Leu Arg Val Asp
165 170 175
Gln Ser Ile Leu Thr Gly Glu Ser Val Ser Val Ile Lys His Thr Asp
180 185 190
Pro Val Pro Asp Pro Arg Ala Val Asn Gln Asp Lys Lys Asn Met Leu
195 200 205
Phe Ser Gly Thr Asn Ile Ala Ala Gly Lys Ala Met Gly Val Val Val
210 215 220
Ala Thr Gly Val Asn Thr Glu Ile Gly Lys Ile Arg Asp Glu Met Val
225 230 235 240
Ala Thr Glu Gln Glu Arg Thr Pro Leu Gln Gln Lys Leu Asp Glu Phe
245 250 255
Gly Glu Gln Leu Ser Lys Val Ile Ser Leu Ile Cys Ile Ala Val Trp
260 265 270
Ile Ile Asn Ile Gly His Phe Asn Asp Pro Val His Gly Gly Ser Trp
275 280 285
Ile Arg Gly Ala Ile Tyr Tyr Phe Lys Ile Ala Val Ala Leu Ala Val
290 295 300
Ala Ala Ile Pro Glu Gly Leu Pro Ala Val Ile Thr Thr Cys Leu Ala
305 310 315 320
Leu Gly Thr Arg Arg Met Ala Lys Lys Asn Ala Ile Val Arg Ser Leu
325 330 335
Pro Ser Val Glu Thr Leu Gly Cys Thr Ser Val Ile Cys Ser Asp Lys
340 345 350
Thr Gly Thr Leu Thr Thr Asn Gln Met Ser Val Cys Arg Met Phe Ile
355 360 365
Leu Asp Arg Val Glu Gly Asp Thr Cys Ser Leu Asn Glu Phe Thr Ile
370 375 380
Thr Gly Ser Thr Tyr Ala Pro Ile Gly Glu Val His Lys Asp Asp Lys
385 390 395 400
Pro Val Asn Cys His Gln Tyr Asp Gly Leu Val Glu Leu Ala Thr Ile
405 410 415
Cys Ala Leu Cys Asn Asp Ser Ala Leu Asp Tyr Asn Glu Ala Lys Gly
420 425 430
Val Tyr Glu Lys Val Gly Glu Ala Thr Glu Thr Ala Leu Thr Cys Leu
435 440 445
Val Glu Lys Met Asn Val Phe Asp Thr Glu Leu Lys Gly Leu Ser Lys
450 455 460
Ile Glu Arg Ala Asn Ala Cys Asn Ser Val Ile Lys Gln Leu Met Lys
465 470 475 480
Lys Glu Phe Thr Leu Glu Phe Ser Arg Asp Arg Lys Ser Met Ser Val
485 490 495
Tyr Cys Thr Pro Asn Lys Pro Ser Arg Thr Ser Met Ser Lys Met Phe
500 505 510
Val Lys Gly Ala Pro Glu Gly Val Ile Asp Arg Cys Thr His Ile Arg
515 520 525
Val Gly Ser Thr Lys Val Pro Met Thr Ser Gly Val Lys Gln Lys Ile
530 535 540
Met Ser Val Ile Arg Glu Trp Gly Ser Gly Ser Asp Thr Leu Arg Cys
545 550 555 560
Leu Ala Leu Ala Thr His Asp Asn Pro Leu Arg Arg Glu Glu Met His
565 570 575
Leu Glu Asp Ser Ala Asn Phe Ile Lys Tyr Glu Thr Asn Leu Thr Phe
580 585 590
Val Gly Cys Val Gly Met Leu Asp Pro Pro Arg Ile Glu Val Ala Ser
595 600 605
Ser Val Lys Leu Cys Arg Gln Ala Gly Ile Arg Val Ile Met Ile Thr
610 615 620
Gly Asp Asn Lys Gly Thr Ala Val Ala Ile Cys Arg Arg Ile Gly Ile
625 630 635 640
Phe Gly Gln Asp Glu Asp Val Thr Ser Lys Ala Phe Thr Gly Arg Glu
645 650 655
Phe Asp Glu Leu Asn Pro Ser Ala Gln Arg Asp Ala Cys Leu Asn Ala
660 665 670
Arg Cys Phe Ala Arg Val Glu Pro Ser His Lys Ser Lys Ile Val Glu
675 680 685
Phe Leu Gln Ser Phe Asp Glu Ile Thr Ala Met Thr Gly Asp Gly Val
690 695 700
Asn Asp Ala Pro Ala Leu Lys Lys Ala Glu Ile Gly Ile Ala Met Gly
705 710 715 720
Ser Gly Thr Ala Val Ala Lys Thr Ala Ser Glu Met Val Leu Ala Asp
725 730 735
Asp Asn Phe Ser Thr Ile Val Ala Ala Val Glu Glu Gly Arg Ala Ile
740 745 750
Tyr Asn Asn Met Lys Gln Phe Ile Arg Tyr Leu Ile Ser Ser Asn Val
755 760 765
Gly Glu Val Val Cys Ile Phe Leu Thr Ala Ala Leu Gly Phe Pro Glu
770 775 780
Ala Leu Ile Pro Val Gln Leu Leu Trp Val Asn Leu Val Thr Asp Gly
785 790 795 800
Leu Pro Ala Thr Ala Leu Gly Phe Asn Pro Pro Asp Leu Asp Ile Met
805 810 815
Asn Lys Pro Pro Arg Asn Pro Lys Glu Pro Leu Ile Ser Gly Trp Leu
820 825 830
Phe Phe Arg Tyr Leu Ala Ile Gly Cys Tyr Val Gly Ala Ala Thr Val
835 840 845
Gly Ala Ala Ala Trp Trp Phe Ile Ala Ala Asp Gly Gly Pro Arg Val
850 855 860
Ser Phe Tyr Gln Leu Ser His Phe Leu Gln Cys Lys Glu Asp Asn Pro
865 870 875 880
Asp Phe Glu Gly Val Asp Cys Ala Ile Phe Glu Ser Pro Tyr Pro Met
885 890 895
Thr Met Ala Leu Ser Val Leu Val Thr Ile Glu Met Cys Asn Ala Leu
900 905 910
Asn Ser Leu Ser Glu Asn Gln Ser Leu Leu Arg Met Pro Pro Trp Glu
915 920 925
Asn Ile Trp Leu Val Gly Ser Ile Cys Leu Ser Met Ser Leu His Phe
930 935 940
Leu Ile Leu Tyr Val Glu Pro Leu Pro Leu Ile Phe Gln Ile Thr Pro
945 950 955 960
Leu Asn Val Thr Gln Trp Leu Met Val Leu Lys Ile Ser Leu Pro Val
965 970 975
Ile Leu Met Asp Glu Thr Leu Lys Phe Val Ala Arg Asn Tyr Leu Glu
980 985 990
Pro Gly Lys Glu Cys Val Gln Pro Ala Thr Lys Ser Cys Ser Phe Ser
995 1000 1005
Ala Cys Thr Asp Gly Ile Ser Trp Pro Phe Val Leu Leu Ile Met
1010 1015 1020
Pro Leu Val Ile Trp Val Tyr Ser Thr Asp Thr Asn Phe Ser Asp
1025 1030 1035
Met Phe Trp Ser
1040
<210> 69
<211> 1938
<212> ДНК
<213> Homo sapiens
<220>
<221> misc_feature
<223> hPDIA4
<400> 69
atgaggcccc ggaaagcctt cctgctcctg ctgctcttgg ggctggtgca gctgctggcc 60
gtggcgggtg ccgagggccc ggacgaggat tcttctaaca gagaaaatgc cattgaggat 120
gaagaggagg aggaggagga agatgatgat gaggaagaag acgacttgga agttaaggaa 180
gaaaatggag tcttggtcct aaatgatgca aactttgata attttgtggc tgacaaagac 240
acagtgctgc tggagtttta tgctccatgg tgtggacatt gcaagcagtt tgctccggaa 300
tatgaaaaaa ttgccaacat attaaaggat aaagatcctc ccattcctgt tgccaagatc 360
gatgcaacct cagcgtctgt gctggccagc aggtttgatg tgagtggcta ccccaccatc 420
aagatcctta agaaggggca ggctgtagac tacgagggct ccagaaccca ggaagaaatt 480
gttgccaagg tcagagaagt ctcccagccc gactggacgc ctccaccaga agtcacgctt 540
gtgttgacca aagagaactt tgatgaagtt gtgaatgatg cagatatcat tctggtggag 600
ttttatgccc catggtgtgg acactgcaag aaacttgccc ccgagtatga gaaggccgcc 660
aaggagctca gcaagcgttc tcctccaatt cccctggcaa aggtcgacgc caccgcagaa 720
acagacctgg ccaagaggtt tgatgtctct ggctatccca ccctgaaaat tttccgcaaa 780
ggaaggcctt atgactacaa cggcccacga gaaaaatatg gaatcgttga ttacatgatc 840
gagcagtccg ggcctccctc caaggagatt ctgaccctga agcaggtcca ggagttcctg 900
aaggatggag acgatgtcat catcatcggg gtctttaagg gggagagtga cccagcctac 960
cagcaatacc aggatgccgc taacaacctg agagaagatt acaaatttca ccacactttc 1020
agcacagaaa tagcaaagtt cttgaaagtc tcccaggggc agttggttgt aatgcagcct 1080
gagaaattcc agtccaagta tgagccccgg agccacatga tggacgtcca gggctccacc 1140
caggactcgg ccatcaagga cttcgtgctg aagtacgccc tgcccctggt tggccaccgc 1200
aaggtgtcaa acgatgctaa gcgctacacc aggcgccccc tggtggtcgt ctactacagt 1260
gtggacttca gctttgatta cagagctgca actcagtttt ggcggagcaa agtcctagag 1320
gtggccaagg acttccctga gtacaccttt gccattgcgg acgaagagga ctatgctggg 1380
gaggtgaagg acctggggct cagcgagagt ggggaggatg tcaatgccgc catcctggac 1440
gagagtggga agaagttcgc catggagcca gaggagtttg actctgacac cctccgcgag 1500
tttgtcactg ctttcaaaaa aggaaaactg aagccagtca tcaaatccca gccagtgccc 1560
aagaacaaca agggacccgt caaggtcgtg gtgggaaaga cctttgactc cattgtgatg 1620
gaccccaaga aggacgtcct catcgagttc tacgcgccat ggtgcgggca ctgcaagcag 1680
ctagagcccg tgtacaacag cctggccaag aagtacaagg gccaaaaggg cctggtcatc 1740
gccaagatgg acgccactgc caacgacgtc cccagcgacc gctataaggt ggagggcttc 1800
cccaccatct acttcgcccc cagtggggac aaaaagaacc cagttaaatt tgagggtgga 1860
gacagagatc tggagcattt gagcaagttt atagaagaac atgccacaaa actgagcagg 1920
accaaggaag agctttga 1938
<210> 70
<211> 645
<212> ПРТ
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<223> hPDIA4
<400> 70
Met Arg Pro Arg Lys Ala Phe Leu Leu Leu Leu Leu Leu Gly Leu Val
1 5 10 15
Gln Leu Leu Ala Val Ala Gly Ala Glu Gly Pro Asp Glu Asp Ser Ser
20 25 30
Asn Arg Glu Asn Ala Ile Glu Asp Glu Glu Glu Glu Glu Glu Glu Asp
35 40 45
Asp Asp Glu Glu Glu Asp Asp Leu Glu Val Lys Glu Glu Asn Gly Val
50 55 60
Leu Val Leu Asn Asp Ala Asn Phe Asp Asn Phe Val Ala Asp Lys Asp
65 70 75 80
Thr Val Leu Leu Glu Phe Tyr Ala Pro Trp Cys Gly His Cys Lys Gln
85 90 95
Phe Ala Pro Glu Tyr Glu Lys Ile Ala Asn Ile Leu Lys Asp Lys Asp
100 105 110
Pro Pro Ile Pro Val Ala Lys Ile Asp Ala Thr Ser Ala Ser Val Leu
115 120 125
Ala Ser Arg Phe Asp Val Ser Gly Tyr Pro Thr Ile Lys Ile Leu Lys
130 135 140
Lys Gly Gln Ala Val Asp Tyr Glu Gly Ser Arg Thr Gln Glu Glu Ile
145 150 155 160
Val Ala Lys Val Arg Glu Val Ser Gln Pro Asp Trp Thr Pro Pro Pro
165 170 175
Glu Val Thr Leu Val Leu Thr Lys Glu Asn Phe Asp Glu Val Val Asn
180 185 190
Asp Ala Asp Ile Ile Leu Val Glu Phe Tyr Ala Pro Trp Cys Gly His
195 200 205
Cys Lys Lys Leu Ala Pro Glu Tyr Glu Lys Ala Ala Lys Glu Leu Ser
210 215 220
Lys Arg Ser Pro Pro Ile Pro Leu Ala Lys Val Asp Ala Thr Ala Glu
225 230 235 240
Thr Asp Leu Ala Lys Arg Phe Asp Val Ser Gly Tyr Pro Thr Leu Lys
245 250 255
Ile Phe Arg Lys Gly Arg Pro Tyr Asp Tyr Asn Gly Pro Arg Glu Lys
260 265 270
Tyr Gly Ile Val Asp Tyr Met Ile Glu Gln Ser Gly Pro Pro Ser Lys
275 280 285
Glu Ile Leu Thr Leu Lys Gln Val Gln Glu Phe Leu Lys Asp Gly Asp
290 295 300
Asp Val Ile Ile Ile Gly Val Phe Lys Gly Glu Ser Asp Pro Ala Tyr
305 310 315 320
Gln Gln Tyr Gln Asp Ala Ala Asn Asn Leu Arg Glu Asp Tyr Lys Phe
325 330 335
His His Thr Phe Ser Thr Glu Ile Ala Lys Phe Leu Lys Val Ser Gln
340 345 350
Gly Gln Leu Val Val Met Gln Pro Glu Lys Phe Gln Ser Lys Tyr Glu
355 360 365
Pro Arg Ser His Met Met Asp Val Gln Gly Ser Thr Gln Asp Ser Ala
370 375 380
Ile Lys Asp Phe Val Leu Lys Tyr Ala Leu Pro Leu Val Gly His Arg
385 390 395 400
Lys Val Ser Asn Asp Ala Lys Arg Tyr Thr Arg Arg Pro Leu Val Val
405 410 415
Val Tyr Tyr Ser Val Asp Phe Ser Phe Asp Tyr Arg Ala Ala Thr Gln
420 425 430
Phe Trp Arg Ser Lys Val Leu Glu Val Ala Lys Asp Phe Pro Glu Tyr
435 440 445
Thr Phe Ala Ile Ala Asp Glu Glu Asp Tyr Ala Gly Glu Val Lys Asp
450 455 460
Leu Gly Leu Ser Glu Ser Gly Glu Asp Val Asn Ala Ala Ile Leu Asp
465 470 475 480
Glu Ser Gly Lys Lys Phe Ala Met Glu Pro Glu Glu Phe Asp Ser Asp
485 490 495
Thr Leu Arg Glu Phe Val Thr Ala Phe Lys Lys Gly Lys Leu Lys Pro
500 505 510
Val Ile Lys Ser Gln Pro Val Pro Lys Asn Asn Lys Gly Pro Val Lys
515 520 525
Val Val Val Gly Lys Thr Phe Asp Ser Ile Val Met Asp Pro Lys Lys
530 535 540
Asp Val Leu Ile Glu Phe Tyr Ala Pro Trp Cys Gly His Cys Lys Gln
545 550 555 560
Leu Glu Pro Val Tyr Asn Ser Leu Ala Lys Lys Tyr Lys Gly Gln Lys
565 570 575
Gly Leu Val Ile Ala Lys Met Asp Ala Thr Ala Asn Asp Val Pro Ser
580 585 590
Asp Arg Tyr Lys Val Glu Gly Phe Pro Thr Ile Tyr Phe Ala Pro Ser
595 600 605
Gly Asp Lys Lys Asn Pro Val Lys Phe Glu Gly Gly Asp Arg Asp Leu
610 615 620
Glu His Leu Ser Lys Phe Ile Glu Glu His Ala Thr Lys Leu Ser Arg
625 630 635 640
Thr Lys Glu Glu Leu
645
<210> 71
<211> 1941
<212> ДНК
<213> Homo sapiens
<220>
<221> misc_feature
<223> hHSC70 /HSPA8
<400> 71
atgtccaagg gacctgcagt tggtattgat cttggcacca cctactcttg tgtgggtgtt 60
ttccagcacg gaaaagtcga gataattgcc aatgatcagg gaaaccgaac cactccaagc 120
tatgtcgcct ttacggacac tgaacggttg atcggtgatg ccgcaaagaa tcaagttgca 180
atgaacccca ccaacacagt ttttgatgcc aaacgtctga ttggacgcag atttgatgat 240
gctgttgtcc agtctgatat gaaacattgg ccctttatgg tggtgaatga tgctggcagg 300
cccaaggtcc aagtagaata caagggagag accaaaagct tctatccaga ggaggtgtct 360
tctatggttc tgacaaagat gaaggaaatt gcagaagcct accttgggaa gactgttacc 420
aatgctgtgg tcacagtgcc agcttacttt aatgactctc agcgtcaggc taccaaagat 480
gctggaacta ttgctggtct caatgtactt agaattatta atgagccaac tgctgctgct 540
attgcttacg gcttagacaa aaaggttgga gcagaaagaa acgtgctcat ctttgacctg 600
ggaggtggca cttttgatgt gtcaatcctc actattgagg atggaatctt tgaggtcaag 660
tctacagctg gagacaccca cttgggtgga gaagattttg acaaccgaat ggtcaaccat 720
tttattgctg agtttaagcg caagcataag aaggacatca gtgagaacaa gagagctgta 780
agacgcctcc gtactgcttg tgaacgtgct aagcgtaccc tctcttccag cacccaggcc 840
agtattgaga tcgattctct ctatgaagga atcgacttct atacctccat tacccgtgcc 900
cgatttgaag aactgaatgc tgacctgttc cgtggcaccc tggacccagt agagaaagcc 960
cttcgagatg ccaaactaga caagtcacag attcatgata ttgtcctggt tggtggttct 1020
actcgtatcc ccaagattca gaagcttctc caagacttct tcaatggaaa agaactgaat 1080
aagagcatca accctgatga agctgttgct tatggtgcag ctgtccaggc agccatcttg 1140
tctggagaca agtctgagaa tgttcaagat ttgctgctct tggatgtcac tcctctttcc 1200
cttggtattg aaactgctgg tggagtcatg actgtcctca tcaagcgtaa taccaccatt 1260
cctaccaagc agacacagac cttcactacc tattctgaca accagcctgg tgtgcttatt 1320
caggtttatg aaggcgagcg tgccatgaca aaggataaca acctgcttgg caagtttgaa 1380
ctcacaggca tacctcctgc accccgaggt gttcctcaga ttgaagtcac ttttgacatt 1440
gatgccaatg gtatactcaa tgtctctgct gtggacaaga gtacgggaaa agagaacaag 1500
attactatca ctaatgacaa gggccgtttg agcaaggaag acattgaacg tatggtccag 1560
gaagctgaga agtacaaagc tgaagatgag aagcagaggg acaaggtgtc atccaagaat 1620
tcacttgagt cctatgcctt caacatgaaa gcaactgttg aagatgagaa acttcaaggc 1680
aagattaacg atgaggacaa acagaagatt ctggacaagt gtaatgaaat tatcaactgg 1740
cttgataaga atcagactgc tgagaaggaa gaatttgaac atcaacagaa agagctggag 1800
aaagtttgca accccatcat caccaagctg taccagagtg caggaggcat gccaggagga 1860
atgcctgggg gatttcctgg tggtggagct cctccctctg gtggtgcttc ctcagggccc 1920
accattgaag aggttgatta a 1941
<210> 72
<211> 646
<212> ПРТ
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<223> hHSC70 /HSPA8
<400> 72
Met Ser Lys Gly Pro Ala Val Gly Ile Asp Leu Gly Thr Thr Tyr Ser
1 5 10 15
Cys Val Gly Val Phe Gln His Gly Lys Val Glu Ile Ile Ala Asn Asp
20 25 30
Gln Gly Asn Arg Thr Thr Pro Ser Tyr Val Ala Phe Thr Asp Thr Glu
35 40 45
Arg Leu Ile Gly Asp Ala Ala Lys Asn Gln Val Ala Met Asn Pro Thr
50 55 60
Asn Thr Val Phe Asp Ala Lys Arg Leu Ile Gly Arg Arg Phe Asp Asp
65 70 75 80
Ala Val Val Gln Ser Asp Met Lys His Trp Pro Phe Met Val Val Asn
85 90 95
Asp Ala Gly Arg Pro Lys Val Gln Val Glu Tyr Lys Gly Glu Thr Lys
100 105 110
Ser Phe Tyr Pro Glu Glu Val Ser Ser Met Val Leu Thr Lys Met Lys
115 120 125
Glu Ile Ala Glu Ala Tyr Leu Gly Lys Thr Val Thr Asn Ala Val Val
130 135 140
Thr Val Pro Ala Tyr Phe Asn Asp Ser Gln Arg Gln Ala Thr Lys Asp
145 150 155 160
Ala Gly Thr Ile Ala Gly Leu Asn Val Leu Arg Ile Ile Asn Glu Pro
165 170 175
Thr Ala Ala Ala Ile Ala Tyr Gly Leu Asp Lys Lys Val Gly Ala Glu
180 185 190
Arg Asn Val Leu Ile Phe Asp Leu Gly Gly Gly Thr Phe Asp Val Ser
195 200 205
Ile Leu Thr Ile Glu Asp Gly Ile Phe Glu Val Lys Ser Thr Ala Gly
210 215 220
Asp Thr His Leu Gly Gly Glu Asp Phe Asp Asn Arg Met Val Asn His
225 230 235 240
Phe Ile Ala Glu Phe Lys Arg Lys His Lys Lys Asp Ile Ser Glu Asn
245 250 255
Lys Arg Ala Val Arg Arg Leu Arg Thr Ala Cys Glu Arg Ala Lys Arg
260 265 270
Thr Leu Ser Ser Ser Thr Gln Ala Ser Ile Glu Ile Asp Ser Leu Tyr
275 280 285
Glu Gly Ile Asp Phe Tyr Thr Ser Ile Thr Arg Ala Arg Phe Glu Glu
290 295 300
Leu Asn Ala Asp Leu Phe Arg Gly Thr Leu Asp Pro Val Glu Lys Ala
305 310 315 320
Leu Arg Asp Ala Lys Leu Asp Lys Ser Gln Ile His Asp Ile Val Leu
325 330 335
Val Gly Gly Ser Thr Arg Ile Pro Lys Ile Gln Lys Leu Leu Gln Asp
340 345 350
Phe Phe Asn Gly Lys Glu Leu Asn Lys Ser Ile Asn Pro Asp Glu Ala
355 360 365
Val Ala Tyr Gly Ala Ala Val Gln Ala Ala Ile Leu Ser Gly Asp Lys
370 375 380
Ser Glu Asn Val Gln Asp Leu Leu Leu Leu Asp Val Thr Pro Leu Ser
385 390 395 400
Leu Gly Ile Glu Thr Ala Gly Gly Val Met Thr Val Leu Ile Lys Arg
405 410 415
Asn Thr Thr Ile Pro Thr Lys Gln Thr Gln Thr Phe Thr Thr Tyr Ser
420 425 430
Asp Asn Gln Pro Gly Val Leu Ile Gln Val Tyr Glu Gly Glu Arg Ala
435 440 445
Met Thr Lys Asp Asn Asn Leu Leu Gly Lys Phe Glu Leu Thr Gly Ile
450 455 460
Pro Pro Ala Pro Arg Gly Val Pro Gln Ile Glu Val Thr Phe Asp Ile
465 470 475 480
Asp Ala Asn Gly Ile Leu Asn Val Ser Ala Val Asp Lys Ser Thr Gly
485 490 495
Lys Glu Asn Lys Ile Thr Ile Thr Asn Asp Lys Gly Arg Leu Ser Lys
500 505 510
Glu Asp Ile Glu Arg Met Val Gln Glu Ala Glu Lys Tyr Lys Ala Glu
515 520 525
Asp Glu Lys Gln Arg Asp Lys Val Ser Ser Lys Asn Ser Leu Glu Ser
530 535 540
Tyr Ala Phe Asn Met Lys Ala Thr Val Glu Asp Glu Lys Leu Gln Gly
545 550 555 560
Lys Ile Asn Asp Glu Asp Lys Gln Lys Ile Leu Asp Lys Cys Asn Glu
565 570 575
Ile Ile Asn Trp Leu Asp Lys Asn Gln Thr Ala Glu Lys Glu Glu Phe
580 585 590
Glu His Gln Gln Lys Glu Leu Glu Lys Val Cys Asn Pro Ile Ile Thr
595 600 605
Lys Leu Tyr Gln Ser Ala Gly Gly Met Pro Gly Gly Met Pro Gly Gly
610 615 620
Phe Pro Gly Gly Gly Ala Pro Pro Ser Gly Gly Ala Ser Ser Gly Pro
625 630 635 640
Thr Ile Glu Glu Val Asp
645
<210> 73
<211> 3000
<212> ДНК
<213> Homo sapiens
<220>
<221> misc_feature
<223> hHYOU1
<400> 73
atggcagaca aagttaggag gcagaggccg aggaggcgag tctgttgggc cttggtggct 60
gtgctcttgg cagacctgtt ggcactgagt gatacactgg cagtgatgtc tgtggacctg 120
ggcagtgagt ccatgaaggt ggccattgtc aaacctggag tgcccatgga aattgtcttg 180
aataaggaat ctcggaggaa aacaccggtg atcgtgaccc tgaaagaaaa tgaaagattc 240
tttggagaca gtgcagcaag catggcgatt aagaatccaa aggctacgct acgttacttc 300
cagcacctcc tggggaagca ggcagataac ccccatgtag ctctttacca ggcccgcttc 360
ccggagcacg agctgacttt cgacccacag aggcagactg tgcactttca gatcagctcg 420
cagctgcagt tctcacctga ggaagtgttg ggcatggttc tcaattattc tcgttctcta 480
gctgaagatt ttgcagagca gcccatcaag gatgcagtga tcaccgtgcc agtcttcttc 540
aaccaggccg agcgccgagc tgtgctgcag gctgctcgta tggctggcct caaagtgctg 600
cagctcatca atgacaacac cgccactgcc ctcagctatg gtgtcttccg ccggaaagat 660
attaacacca ctgcccagaa tatcatgttc tatgacatgg gctcaggcag caccgtatgc 720
accattgtga cctaccagat ggtgaagact aaggaagctg ggatgcagcc acagctgcag 780
atccggggag taggatttga ccgtaccctg gggggcctgg agatggagct ccggcttcga 840
gaacgcctgg ctgggctttt caatgagcag cgcaagggtc agagagcaaa ggatgtgcgg 900
gagaacccgc gtgccatggc caagctgctg cgtgaggcta atcggctcaa aaccgtcctc 960
agtgccaacg ctgaccacat ggcacagatt gaaggcctga tggatgatgt ggacttcaag 1020
gcaaaagtga ctcgtgtgga atttgaggag ttgtgtgcag acttgtttga gcgggtgcct 1080
gggcctgtac agcaggccct ccagagtgcc gaaatgagtc tggatgagat tgagcaggtg 1140
atcctggtgg gtggggccac tcgggtcccc agagttcagg aggtgctgct gaaggccgtg 1200
ggcaaggagg agctggggaa gaacatcaat gcagatgaag cagccgccat gggggcagtg 1260
taccaggcag ctgcgctcag caaagccttt aaagtgaagc catttgtcgt ccgagatgca 1320
gtggtctacc ccatcctggt ggagttcacg agggaggtgg aggaggagcc tgggattcac 1380
agcctgaagc acaataaacg ggtactcttc tctcggatgg ggccctaccc tcaacgcaaa 1440
gtcatcacct ttaaccgcta cagccatgat ttcaacttcc acatcaacta cggcgacctg 1500
ggcttcctgg ggcctgaaga tcttcgggta tttggctccc agaatctgac cacagtgaag 1560
ctaaaagggg tgggtgacag cttcaagaag tatcctgact acgagtccaa gggcatcaag 1620
gctcacttca acctggatga gagtggcgtg ctcagtctag acagggtgga gtctgtattt 1680
gagacactgg tagaggacag cgcagaagag gaatctactc tcaccaaact tggcaacacc 1740
atttccagcc tgtttggagg cggtaccaca ccagatgcca aggagaatgg tactgatact 1800
gtccaggagg aagaggagag ccctgcagag gggagcaagg acgagcctgg ggagcaggtg 1860
gagctcaagg aggaagctga ggccccagtg gaggatggct ctcagccccc accccctgaa 1920
cctaagggag atgcaacccc tgagggagaa aaggccacag aaaaagaaaa tggggacaag 1980
tctgaggccc agaaaccaag tgagaaggca gaggcagggc ctgagggcgt cgctccagcc 2040
ccagagggag agaagaagca gaagcccgcc aggaagcggc gaatggtaga ggagatcggg 2100
gtggagctgg ttgttctgga cctgcctgac ttgccagagg ataagctggc tcagtcggtg 2160
cagaaacttc aggacttgac actccgagac ctggagaagc aggaacggga aaaagctgcc 2220
aacagcttgg aagcattcat atttgagacc caggacaagc tgtaccagcc cgagtaccag 2280
gaagtgtcca cagaggagca gcgtgaggag atctctggga agctcagcgc cgcatccacc 2340
tggctggagg atgagggtgt tggagccacc acagtgatgt tgaaggagaa gctggctgag 2400
ctgaggaagc tgtgccaagg gctgtttttt cgggtagagg agcgcaagaa gtggcccgaa 2460
cggctgtctg ccctcgataa tctcctcaac cattccagca tgttcctcaa gggggcccgg 2520
ctcatcccag agatggacca gatcttcact gaggtggaga tgacaacgtt agagaaagtc 2580
atcaatgaga cctgggcctg gaagaatgca actctggccg agcaggctaa gctgcccgcc 2640
acagagaagc ctgtgttgct ctcaaaagac attgaagcta agatgatggc cctggaccga 2700
gaggtgcagt atctgctcaa taaggccaag tttaccaagc cccggccccg gcctaaggac 2760
aagaatggga cccgggcaga gccacccctc aatgccagtg ccagtgacca gggggagaag 2820
gtcatccctc cagcaggcca gactgaagat gcagagccca tttcagaacc tgagaaagta 2880
gagactggat ccgagccagg agacactgag cctttggagt taggaggtcc tggagcagaa 2940
cctgaacaga aagaacaatc gacaggacag aagcggcctt tgaagaacga cgaactataa 3000
<210> 74
<211> 999
<212> ПРТ
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<223> hHYOU1
<400> 74
Met Ala Asp Lys Val Arg Arg Gln Arg Pro Arg Arg Arg Val Cys Trp
1 5 10 15
Ala Leu Val Ala Val Leu Leu Ala Asp Leu Leu Ala Leu Ser Asp Thr
20 25 30
Leu Ala Val Met Ser Val Asp Leu Gly Ser Glu Ser Met Lys Val Ala
35 40 45
Ile Val Lys Pro Gly Val Pro Met Glu Ile Val Leu Asn Lys Glu Ser
50 55 60
Arg Arg Lys Thr Pro Val Ile Val Thr Leu Lys Glu Asn Glu Arg Phe
65 70 75 80
Phe Gly Asp Ser Ala Ala Ser Met Ala Ile Lys Asn Pro Lys Ala Thr
85 90 95
Leu Arg Tyr Phe Gln His Leu Leu Gly Lys Gln Ala Asp Asn Pro His
100 105 110
Val Ala Leu Tyr Gln Ala Arg Phe Pro Glu His Glu Leu Thr Phe Asp
115 120 125
Pro Gln Arg Gln Thr Val His Phe Gln Ile Ser Ser Gln Leu Gln Phe
130 135 140
Ser Pro Glu Glu Val Leu Gly Met Val Leu Asn Tyr Ser Arg Ser Leu
145 150 155 160
Ala Glu Asp Phe Ala Glu Gln Pro Ile Lys Asp Ala Val Ile Thr Val
165 170 175
Pro Val Phe Phe Asn Gln Ala Glu Arg Arg Ala Val Leu Gln Ala Ala
180 185 190
Arg Met Ala Gly Leu Lys Val Leu Gln Leu Ile Asn Asp Asn Thr Ala
195 200 205
Thr Ala Leu Ser Tyr Gly Val Phe Arg Arg Lys Asp Ile Asn Thr Thr
210 215 220
Ala Gln Asn Ile Met Phe Tyr Asp Met Gly Ser Gly Ser Thr Val Cys
225 230 235 240
Thr Ile Val Thr Tyr Gln Met Val Lys Thr Lys Glu Ala Gly Met Gln
245 250 255
Pro Gln Leu Gln Ile Arg Gly Val Gly Phe Asp Arg Thr Leu Gly Gly
260 265 270
Leu Glu Met Glu Leu Arg Leu Arg Glu Arg Leu Ala Gly Leu Phe Asn
275 280 285
Glu Gln Arg Lys Gly Gln Arg Ala Lys Asp Val Arg Glu Asn Pro Arg
290 295 300
Ala Met Ala Lys Leu Leu Arg Glu Ala Asn Arg Leu Lys Thr Val Leu
305 310 315 320
Ser Ala Asn Ala Asp His Met Ala Gln Ile Glu Gly Leu Met Asp Asp
325 330 335
Val Asp Phe Lys Ala Lys Val Thr Arg Val Glu Phe Glu Glu Leu Cys
340 345 350
Ala Asp Leu Phe Glu Arg Val Pro Gly Pro Val Gln Gln Ala Leu Gln
355 360 365
Ser Ala Glu Met Ser Leu Asp Glu Ile Glu Gln Val Ile Leu Val Gly
370 375 380
Gly Ala Thr Arg Val Pro Arg Val Gln Glu Val Leu Leu Lys Ala Val
385 390 395 400
Gly Lys Glu Glu Leu Gly Lys Asn Ile Asn Ala Asp Glu Ala Ala Ala
405 410 415
Met Gly Ala Val Tyr Gln Ala Ala Ala Leu Ser Lys Ala Phe Lys Val
420 425 430
Lys Pro Phe Val Val Arg Asp Ala Val Val Tyr Pro Ile Leu Val Glu
435 440 445
Phe Thr Arg Glu Val Glu Glu Glu Pro Gly Ile His Ser Leu Lys His
450 455 460
Asn Lys Arg Val Leu Phe Ser Arg Met Gly Pro Tyr Pro Gln Arg Lys
465 470 475 480
Val Ile Thr Phe Asn Arg Tyr Ser His Asp Phe Asn Phe His Ile Asn
485 490 495
Tyr Gly Asp Leu Gly Phe Leu Gly Pro Glu Asp Leu Arg Val Phe Gly
500 505 510
Ser Gln Asn Leu Thr Thr Val Lys Leu Lys Gly Val Gly Asp Ser Phe
515 520 525
Lys Lys Tyr Pro Asp Tyr Glu Ser Lys Gly Ile Lys Ala His Phe Asn
530 535 540
Leu Asp Glu Ser Gly Val Leu Ser Leu Asp Arg Val Glu Ser Val Phe
545 550 555 560
Glu Thr Leu Val Glu Asp Ser Ala Glu Glu Glu Ser Thr Leu Thr Lys
565 570 575
Leu Gly Asn Thr Ile Ser Ser Leu Phe Gly Gly Gly Thr Thr Pro Asp
580 585 590
Ala Lys Glu Asn Gly Thr Asp Thr Val Gln Glu Glu Glu Glu Ser Pro
595 600 605
Ala Glu Gly Ser Lys Asp Glu Pro Gly Glu Gln Val Glu Leu Lys Glu
610 615 620
Glu Ala Glu Ala Pro Val Glu Asp Gly Ser Gln Pro Pro Pro Pro Glu
625 630 635 640
Pro Lys Gly Asp Ala Thr Pro Glu Gly Glu Lys Ala Thr Glu Lys Glu
645 650 655
Asn Gly Asp Lys Ser Glu Ala Gln Lys Pro Ser Glu Lys Ala Glu Ala
660 665 670
Gly Pro Glu Gly Val Ala Pro Ala Pro Glu Gly Glu Lys Lys Gln Lys
675 680 685
Pro Ala Arg Lys Arg Arg Met Val Glu Glu Ile Gly Val Glu Leu Val
690 695 700
Val Leu Asp Leu Pro Asp Leu Pro Glu Asp Lys Leu Ala Gln Ser Val
705 710 715 720
Gln Lys Leu Gln Asp Leu Thr Leu Arg Asp Leu Glu Lys Gln Glu Arg
725 730 735
Glu Lys Ala Ala Asn Ser Leu Glu Ala Phe Ile Phe Glu Thr Gln Asp
740 745 750
Lys Leu Tyr Gln Pro Glu Tyr Gln Glu Val Ser Thr Glu Glu Gln Arg
755 760 765
Glu Glu Ile Ser Gly Lys Leu Ser Ala Ala Ser Thr Trp Leu Glu Asp
770 775 780
Glu Gly Val Gly Ala Thr Thr Val Met Leu Lys Glu Lys Leu Ala Glu
785 790 795 800
Leu Arg Lys Leu Cys Gln Gly Leu Phe Phe Arg Val Glu Glu Arg Lys
805 810 815
Lys Trp Pro Glu Arg Leu Ser Ala Leu Asp Asn Leu Leu Asn His Ser
820 825 830
Ser Met Phe Leu Lys Gly Ala Arg Leu Ile Pro Glu Met Asp Gln Ile
835 840 845
Phe Thr Glu Val Glu Met Thr Thr Leu Glu Lys Val Ile Asn Glu Thr
850 855 860
Trp Ala Trp Lys Asn Ala Thr Leu Ala Glu Gln Ala Lys Leu Pro Ala
865 870 875 880
Thr Glu Lys Pro Val Leu Leu Ser Lys Asp Ile Glu Ala Lys Met Met
885 890 895
Ala Leu Asp Arg Glu Val Gln Tyr Leu Leu Asn Lys Ala Lys Phe Thr
900 905 910
Lys Pro Arg Pro Arg Pro Lys Asp Lys Asn Gly Thr Arg Ala Glu Pro
915 920 925
Pro Leu Asn Ala Ser Ala Ser Asp Gln Gly Glu Lys Val Ile Pro Pro
930 935 940
Ala Gly Gln Thr Glu Asp Ala Glu Pro Ile Ser Glu Pro Glu Lys Val
945 950 955 960
Glu Thr Gly Ser Glu Pro Gly Asp Thr Glu Pro Leu Glu Leu Gly Gly
965 970 975
Pro Gly Ala Glu Pro Glu Gln Lys Glu Gln Ser Thr Gly Gln Lys Arg
980 985 990
Pro Leu Lys Asn Asp Glu Leu
995
<210> 75
<211> 1305
<212> ДНК
<213> Homo sapiens
<220>
<221> misc_feature
<223> hCMP-SAS
<400> 75
atggactcgg tggagaaggg ggccgccacc tccgtctcca acccgcgggg gcgaccgtcc 60
cggggccggc cgccgaagct gcagcgcaac tctcgcggcg gccagggccg aggtgtggag 120
aagcccccgc acctggcagc cctaattctg gcccggggag gcagcaaagg catccccctg 180
aagaacatta agcacctggc gggggtcccg ctcattggct gggtcctgcg tgcggccctg 240
gattcagggg ccttccagag tgtatgggtt tcgacagacc atgatgaaat tgagaatgtg 300
gccaaacaat ttggtgcaca agttcatcga agaagttctg aagtttcaaa agacagctct 360
acctcactag atgccatcat agaatttctt aattatcata atgaggttga cattgtagga 420
aatattcaag ctacttctcc atgtttacat cctactgatc ttcaaaaagt tgcagaaatg 480
attcgagaag aaggatatga ttctgttttc tctgttgtga gacgccatca gtttcgatgg 540
agtgaaattc agaaaggagt tcgtgaagtg accgaacctc tgaatttaaa tccagctaaa 600
cggcctcgtc gacaagactg ggatggagaa ttatatgaaa atggctcatt ttattttgct 660
aaaagacatt tgatagagat gggttacttg cagggtggaa aaatggcata ctacgaaatg 720
cgagctgaac atagtgtgga tatagatgtg gatattgatt ggcctattgc agagcaaaga 780
gtattaagat atggctattt tggcaaagag aagcttaagg aaataaaact tttggtttgc 840
aatattgatg gatgtctcac caatggccac atttatgtat caggagacca aaaagaaata 900
atatcttatg atgtaaaaga tgctattggg ataagtttat taaagaaaag tggtattgag 960
gtgaggctaa tctcagaaag ggcctgttca aagcagacgc tgtcttcttt aaaactggat 1020
tgcaaaatgg aagtcagtgt atcagacaag ctagcagttg tagatgaatg gagaaaagaa 1080
atgggcctgt gctggaaaga agtggcatat cttggaaatg aagtgtctga tgaagagtgc 1140
ttgaagagag tgggcctaag tggcgctcct gctgatgcct gttctactgc ccagaaggct 1200
gttggataca tttgcaaatg taatggtggc cgtggtgcca tccgagaatt tgcagagcac 1260
atttgcctac taatggaaaa ggttaataat tcatgccaaa aatag 1305
<210> 76
<211> 434
<212> ПРТ
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<223> hCMP-SAS
<400> 76
Met Asp Ser Val Glu Lys Gly Ala Ala Thr Ser Val Ser Asn Pro Arg
1 5 10 15
Gly Arg Pro Ser Arg Gly Arg Pro Pro Lys Leu Gln Arg Asn Ser Arg
20 25 30
Gly Gly Gln Gly Arg Gly Val Glu Lys Pro Pro His Leu Ala Ala Leu
35 40 45
Ile Leu Ala Arg Gly Gly Ser Lys Gly Ile Pro Leu Lys Asn Ile Lys
50 55 60
His Leu Ala Gly Val Pro Leu Ile Gly Trp Val Leu Arg Ala Ala Leu
65 70 75 80
Asp Ser Gly Ala Phe Gln Ser Val Trp Val Ser Thr Asp His Asp Glu
85 90 95
Ile Glu Asn Val Ala Lys Gln Phe Gly Ala Gln Val His Arg Arg Ser
100 105 110
Ser Glu Val Ser Lys Asp Ser Ser Thr Ser Leu Asp Ala Ile Ile Glu
115 120 125
Phe Leu Asn Tyr His Asn Glu Val Asp Ile Val Gly Asn Ile Gln Ala
130 135 140
Thr Ser Pro Cys Leu His Pro Thr Asp Leu Gln Lys Val Ala Glu Met
145 150 155 160
Ile Arg Glu Glu Gly Tyr Asp Ser Val Phe Ser Val Val Arg Arg His
165 170 175
Gln Phe Arg Trp Ser Glu Ile Gln Lys Gly Val Arg Glu Val Thr Glu
180 185 190
Pro Leu Asn Leu Asn Pro Ala Lys Arg Pro Arg Arg Gln Asp Trp Asp
195 200 205
Gly Glu Leu Tyr Glu Asn Gly Ser Phe Tyr Phe Ala Lys Arg His Leu
210 215 220
Ile Glu Met Gly Tyr Leu Gln Gly Gly Lys Met Ala Tyr Tyr Glu Met
225 230 235 240
Arg Ala Glu His Ser Val Asp Ile Asp Val Asp Ile Asp Trp Pro Ile
245 250 255
Ala Glu Gln Arg Val Leu Arg Tyr Gly Tyr Phe Gly Lys Glu Lys Leu
260 265 270
Lys Glu Ile Lys Leu Leu Val Cys Asn Ile Asp Gly Cys Leu Thr Asn
275 280 285
Gly His Ile Tyr Val Ser Gly Asp Gln Lys Glu Ile Ile Ser Tyr Asp
290 295 300
Val Lys Asp Ala Ile Gly Ile Ser Leu Leu Lys Lys Ser Gly Ile Glu
305 310 315 320
Val Arg Leu Ile Ser Glu Arg Ala Cys Ser Lys Gln Thr Leu Ser Ser
325 330 335
Leu Lys Leu Asp Cys Lys Met Glu Val Ser Val Ser Asp Lys Leu Ala
340 345 350
Val Val Asp Glu Trp Arg Lys Glu Met Gly Leu Cys Trp Lys Glu Val
355 360 365
Ala Tyr Leu Gly Asn Glu Val Ser Asp Glu Glu Cys Leu Lys Arg Val
370 375 380
Gly Leu Ser Gly Ala Pro Ala Asp Ala Cys Ser Thr Ala Gln Lys Ala
385 390 395 400
Val Gly Tyr Ile Cys Lys Cys Asn Gly Gly Arg Gly Ala Ile Arg Glu
405 410 415
Phe Ala Glu His Ile Cys Leu Leu Met Glu Lys Val Asn Asn Ser Cys
420 425 430
Gln Lys
<210> 77
<211> 1353
<212> ДНК
<213> Homo sapiens
<220>
<221> misc_feature
<223> hбеклин-1
<400> 77
atggaagggt ctaagacgtc caacaacagc accatgcagg tgagcttcgt gtgccagcgc 60
tgcagccagc ccctgaaact ggacacgagt ttcaagatcc tggaccgtgt caccatccag 120
gaactcacag ctccattact taccacagcc caggcgaaac caggagagac ccaggaggaa 180
gagactaact caggagagga gccatttatt gaaactcctc gccaggatgg tgtctctcgc 240
agattcatcc ccccagccag gatgatgtcc acagaaagtg ccaacagctt cactctgatt 300
ggggaggcat ctgatggcgg caccatggag aacctcagcc gaagactgaa ggtcactggg 360
gacctttttg acatcatgtc gggccagaca gatgtggatc acccactctg tgaggaatgc 420
acagatactc ttttagacca gctggacact cagctcaacg tcactgaaaa tgagtgtcag 480
aactacaaac gctgtttgga gatcttagag caaatgaatg aggatgacag tgaacagtta 540
cagatggagc taaaggagct ggcactagag gaggagaggc tgatccagga gctggaagac 600
gtggaaaaga accgcaagat agtggcagaa aatctcgaga aggtccaggc tgaggctgag 660
agactggatc aggaggaagc tcagtatcag agagaataca gtgaatttaa acgacagcag 720
ctggagctgg atgatgagct gaagagtgtt gaaaaccaga tgcgttatgc ccagacgcag 780
ctggataagc tgaagaaaac caacgtcttt aatgcaacct tccacatctg gcacagtgga 840
cagtttggca caatcaataa cttcaggctg ggtcgcctgc ccagtgttcc cgtggaatgg 900
aatgagatta atgctgcttg gggccagact gtgttgctgc tccatgctct ggccaataag 960
atgggtctga aatttcagag ataccgactt gttccttacg gaaaccattc atatctggag 1020
tctctgacag acaaatctaa ggagctgccg ttatactgtt ctggggggtt gcggtttttc 1080
tgggacaaca agtttgacca tgcaatggtg gctttcctgg actgtgtgca gcagttcaaa 1140
gaagaggttg agaaaggcga gacacgtttt tgtcttccct acaggatgga tgtggagaaa 1200
ggcaagattg aagacacagg aggcagtggc ggctcctatt ccatcaaaac ccagtttaac 1260
tctgaggagc agtggacaaa agctctcaag ttcatgctga cgaatcttaa gtggggtctt 1320
gcttgggtgt cctcacaatt ttataacaaa tga 1353
<210> 78
<211> 450
<212> ПРТ
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<223> hбеклин-1
<400> 78
Met Glu Gly Ser Lys Thr Ser Asn Asn Ser Thr Met Gln Val Ser Phe
1 5 10 15
Val Cys Gln Arg Cys Ser Gln Pro Leu Lys Leu Asp Thr Ser Phe Lys
20 25 30
Ile Leu Asp Arg Val Thr Ile Gln Glu Leu Thr Ala Pro Leu Leu Thr
35 40 45
Thr Ala Gln Ala Lys Pro Gly Glu Thr Gln Glu Glu Glu Thr Asn Ser
50 55 60
Gly Glu Glu Pro Phe Ile Glu Thr Pro Arg Gln Asp Gly Val Ser Arg
65 70 75 80
Arg Phe Ile Pro Pro Ala Arg Met Met Ser Thr Glu Ser Ala Asn Ser
85 90 95
Phe Thr Leu Ile Gly Glu Ala Ser Asp Gly Gly Thr Met Glu Asn Leu
100 105 110
Ser Arg Arg Leu Lys Val Thr Gly Asp Leu Phe Asp Ile Met Ser Gly
115 120 125
Gln Thr Asp Val Asp His Pro Leu Cys Glu Glu Cys Thr Asp Thr Leu
130 135 140
Leu Asp Gln Leu Asp Thr Gln Leu Asn Val Thr Glu Asn Glu Cys Gln
145 150 155 160
Asn Tyr Lys Arg Cys Leu Glu Ile Leu Glu Gln Met Asn Glu Asp Asp
165 170 175
Ser Glu Gln Leu Gln Met Glu Leu Lys Glu Leu Ala Leu Glu Glu Glu
180 185 190
Arg Leu Ile Gln Glu Leu Glu Asp Val Glu Lys Asn Arg Lys Ile Val
195 200 205
Ala Glu Asn Leu Glu Lys Val Gln Ala Glu Ala Glu Arg Leu Asp Gln
210 215 220
Glu Glu Ala Gln Tyr Gln Arg Glu Tyr Ser Glu Phe Lys Arg Gln Gln
225 230 235 240
Leu Glu Leu Asp Asp Glu Leu Lys Ser Val Glu Asn Gln Met Arg Tyr
245 250 255
Ala Gln Thr Gln Leu Asp Lys Leu Lys Lys Thr Asn Val Phe Asn Ala
260 265 270
Thr Phe His Ile Trp His Ser Gly Gln Phe Gly Thr Ile Asn Asn Phe
275 280 285
Arg Leu Gly Arg Leu Pro Ser Val Pro Val Glu Trp Asn Glu Ile Asn
290 295 300
Ala Ala Trp Gly Gln Thr Val Leu Leu Leu His Ala Leu Ala Asn Lys
305 310 315 320
Met Gly Leu Lys Phe Gln Arg Tyr Arg Leu Val Pro Tyr Gly Asn His
325 330 335
Ser Tyr Leu Glu Ser Leu Thr Asp Lys Ser Lys Glu Leu Pro Leu Tyr
340 345 350
Cys Ser Gly Gly Leu Arg Phe Phe Trp Asp Asn Lys Phe Asp His Ala
355 360 365
Met Val Ala Phe Leu Asp Cys Val Gln Gln Phe Lys Glu Glu Val Glu
370 375 380
Lys Gly Glu Thr Arg Phe Cys Leu Pro Tyr Arg Met Asp Val Glu Lys
385 390 395 400
Gly Lys Ile Glu Asp Thr Gly Gly Ser Gly Gly Ser Tyr Ser Ile Lys
405 410 415
Thr Gln Phe Asn Ser Glu Glu Gln Trp Thr Lys Ala Leu Lys Phe Met
420 425 430
Leu Thr Asn Leu Lys Trp Gly Leu Ala Trp Val Ser Ser Gln Phe Tyr
435 440 445
Asn Lys
450
<210> 79
<211> 1077
<212> ДНК
<213> Homo sapiens
<220>
<221> misc_feature
<223> hERdj3
<400> 79
atggctccgc agaacctgag caccttttgc ctgttgctgc tatacctcat cggggcggtg 60
attgccggac gagatttcta taagatcttg ggggtgcctc gaagtgcctc tataaaggat 120
attaaaaagg cctataggaa actagccctg cagcttcatc ccgaccggaa ccctgatgat 180
ccacaagccc aggagaaatt ccaggatctg ggtgctgctt atgaggttct gtcagatagt 240
gagaaacgga aacagtacga tacttatggt gaagaaggat taaaagatgg tcatcagagc 300
tcccatggag acattttttc acacttcttt ggggattttg gtttcatgtt tggaggaacc 360
cctcgtcagc aagacagaaa tattccaaga ggaagtgata ttattgtaga tctagaagtc 420
actttggaag aagtatatgc aggaaatttt gtggaagtag ttagaaacaa acctgtggca 480
aggcaggctc ctggcaaacg gaagtgcaat tgtcggcaag agatgcggac cacccagctg 540
ggccctgggc gcttccaaat gacccaggag gtggtctgcg acgaatgccc taatgtcaaa 600
ctagtgaatg aagaacgaac gctggaagta gaaatagagc ctggggtgag agacggcatg 660
gagtacccct ttattggaga aggtgagcct cacgtggatg gggagcctgg agatttacgg 720
ttccgaatca aagttgtcaa gcacccaata tttgaaagga gaggagatga tttgtacaca 780
aatgtgacaa tctcattagt tgagtcactg gttggctttg agatggatat tactcacttg 840
gatggtcaca aggtacatat ttcccgggat aagatcacca ggccaggagc gaagctatgg 900
aagaaagggg aagggctccc caactttgac aacaacaata tcaagggctc tttgataatc 960
acttttgatg tggattttcc aaaagaacag ttaacagagg aagcgagaga aggtatcaaa 1020
cagctactga aacaagggtc agtgcagaag gtatacaatg gactgcaagg atattga 1077
<210> 80
<211> 358
<212> ПРТ
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<223> hERdj3
<400> 80
Met Ala Pro Gln Asn Leu Ser Thr Phe Cys Leu Leu Leu Leu Tyr Leu
1 5 10 15
Ile Gly Ala Val Ile Ala Gly Arg Asp Phe Tyr Lys Ile Leu Gly Val
20 25 30
Pro Arg Ser Ala Ser Ile Lys Asp Ile Lys Lys Ala Tyr Arg Lys Leu
35 40 45
Ala Leu Gln Leu His Pro Asp Arg Asn Pro Asp Asp Pro Gln Ala Gln
50 55 60
Glu Lys Phe Gln Asp Leu Gly Ala Ala Tyr Glu Val Leu Ser Asp Ser
65 70 75 80
Glu Lys Arg Lys Gln Tyr Asp Thr Tyr Gly Glu Glu Gly Leu Lys Asp
85 90 95
Gly His Gln Ser Ser His Gly Asp Ile Phe Ser His Phe Phe Gly Asp
100 105 110
Phe Gly Phe Met Phe Gly Gly Thr Pro Arg Gln Gln Asp Arg Asn Ile
115 120 125
Pro Arg Gly Ser Asp Ile Ile Val Asp Leu Glu Val Thr Leu Glu Glu
130 135 140
Val Tyr Ala Gly Asn Phe Val Glu Val Val Arg Asn Lys Pro Val Ala
145 150 155 160
Arg Gln Ala Pro Gly Lys Arg Lys Cys Asn Cys Arg Gln Glu Met Arg
165 170 175
Thr Thr Gln Leu Gly Pro Gly Arg Phe Gln Met Thr Gln Glu Val Val
180 185 190
Cys Asp Glu Cys Pro Asn Val Lys Leu Val Asn Glu Glu Arg Thr Leu
195 200 205
Glu Val Glu Ile Glu Pro Gly Val Arg Asp Gly Met Glu Tyr Pro Phe
210 215 220
Ile Gly Glu Gly Glu Pro His Val Asp Gly Glu Pro Gly Asp Leu Arg
225 230 235 240
Phe Arg Ile Lys Val Val Lys His Pro Ile Phe Glu Arg Arg Gly Asp
245 250 255
Asp Leu Tyr Thr Asn Val Thr Ile Ser Leu Val Glu Ser Leu Val Gly
260 265 270
Phe Glu Met Asp Ile Thr His Leu Asp Gly His Lys Val His Ile Ser
275 280 285
Arg Asp Lys Ile Thr Arg Pro Gly Ala Lys Leu Trp Lys Lys Gly Glu
290 295 300
Gly Leu Pro Asn Phe Asp Asn Asn Asn Ile Lys Gly Ser Leu Ile Ile
305 310 315 320
Thr Phe Asp Val Asp Phe Pro Lys Glu Gln Leu Thr Glu Glu Ala Arg
325 330 335
Glu Gly Ile Lys Gln Leu Leu Lys Gln Gly Ser Val Gln Lys Val Tyr
340 345 350
Asn Gly Leu Gln Gly Tyr
355
<210> 81
<211> 1764
<212> ДНК
<213> Cricetulus griseus
<220>
<221> misc_feature
<223> CHO_AGE
<400> 81
atgaaaggaa ttctgtgggg agcgttttgt gggaaagcta atgagctgag gcagcgcctg 60
gagctgaaag aaaagggcag gcacgtggtg ctggaggcca cagagaatga ggtggagggc 120
aaaggcggca tgcttctgag ctcaggacag gcctgttgtg aggagaaggg cctggatgct 180
gaggacggtg gtggggacag caatgacctc agcagcgtca gcgagaccac agacagcaca 240
gatgtcaaag acagctcaga gacctctctg actctgcctc ctagagcctc accctgccct 300
gcaggctttt ggattcttga aactggagct agccaaagac agtttcaggc ctggtttact 360
gcggaggagg gtccgctgcc accttcgcct ccttttggcc ctgaactgac ttgcagatgg 420
agcagtctag aacagctaaa ggacatcttg gcgctcggag cacagcctca ctgggtccac 480
aagctccaag gcttctggtt tgttgaggtt atcttgtcag caaaaagatt acaggacatg 540
gagaaggagc gggcttcact gcaggcctgg aaggatcgtg tggcaaagga acttgacagc 600
gtggtcgctt tctggctgca gcactcccat gatcaggaac acgggggttt cttcacatgc 660
cttgcccgca atgggcaggt ctatgatgac cttaaatata tctggctgca ggggagacag 720
gtatggatgt attgtcgctt gtaccgcaat tttgagcgct tccgccgcgc tgagcttcta 780
gatgctgcaa aagcaggtgg tgagtttttg ctgcgttttg cacgggtggc accgcctgcc 840
aagaagtgtg cctttgtgtt gactcgggac ggccgtccag tgaaggtgca gcggaccatt 900
ttcagcgagt gtttctacac cattgctatg aatgagcttt ggagagtaac aaaggaatca 960
cgttaccaga atgaagctgt ggagatgatg gaccagattg tccactgggt acgggaagac 1020
ccagctgggc taggccggcc tcagctctca gggaccccgg atgcagagtc catggcagtg 1080
cccatgatgc tgatgaatct ggtggaccag ctttccgagg aagatgaggc actgacaaag 1140
aaatatgcag agctatggga ctggtgtgcc cagaggattc ttcagcatgt gcagagggat 1200
ggaaaagcta tactagagaa tgtatctcat gatggcaagg aactcactgg ttgccttgga 1260
agacatcaga acccaggcca cgcaatagag gctggctggt tcctgctcca gtatgcccgc 1320
aagaaaggtg acgccaaact tcgtgcacac atcatcgaca agtttctctt gttgcctttc 1380
cgctctggat gggacgctga gcatgggggc ctcttctact tccaggatgt tgatggtttc 1440
tgccccaccc agctggagtg ggacatgaag ctgtggtggc cacacaatga agccatgatt 1500
tccttcctaa tgggttacag ggacagtggt gacccagcct tgctacaaat cttcaatcag 1560
gtggctgagt acaccttcaa gcattttcat gatcccgaga acggcgaatg gttcggctat 1620
ctgaaccgag agggcaaggt ggccctcaac tttaagggag gcccatttaa aggctgcttc 1680
cacgtgccgc ggtgcctggc catgtgcgag aagatgttgg gagacctgct ccaccgtggg 1740
agcgcccctg ctggctcgaa ataa 1764
<210> 82
<211> 587
<212> ПРТ
<213> Cricetulus griseus
<220>
<221> MISC_FEATURE
<223> CHO_AGE
<400> 82
Met Lys Gly Ile Leu Trp Gly Ala Phe Cys Gly Lys Ala Asn Glu Leu
1 5 10 15
Arg Gln Arg Leu Glu Leu Lys Glu Lys Gly Arg His Val Val Leu Glu
20 25 30
Ala Thr Glu Asn Glu Val Glu Gly Lys Gly Gly Met Leu Leu Ser Ser
35 40 45
Gly Gln Ala Cys Cys Glu Glu Lys Gly Leu Asp Ala Glu Asp Gly Gly
50 55 60
Gly Asp Ser Asn Asp Leu Ser Ser Val Ser Glu Thr Thr Asp Ser Thr
65 70 75 80
Asp Val Lys Asp Ser Ser Glu Thr Ser Leu Thr Leu Pro Pro Arg Ala
85 90 95
Ser Pro Cys Pro Ala Gly Phe Trp Ile Leu Glu Thr Gly Ala Ser Gln
100 105 110
Arg Gln Phe Gln Ala Trp Phe Thr Ala Glu Glu Gly Pro Leu Pro Pro
115 120 125
Ser Pro Pro Phe Gly Pro Glu Leu Thr Cys Arg Trp Ser Ser Leu Glu
130 135 140
Gln Leu Lys Asp Ile Leu Ala Leu Gly Ala Gln Pro His Trp Val His
145 150 155 160
Lys Leu Gln Gly Phe Trp Phe Val Glu Val Ile Leu Ser Ala Lys Arg
165 170 175
Leu Gln Asp Met Glu Lys Glu Arg Ala Ser Leu Gln Ala Trp Lys Asp
180 185 190
Arg Val Ala Lys Glu Leu Asp Ser Val Val Ala Phe Trp Leu Gln His
195 200 205
Ser His Asp Gln Glu His Gly Gly Phe Phe Thr Cys Leu Ala Arg Asn
210 215 220
Gly Gln Val Tyr Asp Asp Leu Lys Tyr Ile Trp Leu Gln Gly Arg Gln
225 230 235 240
Val Trp Met Tyr Cys Arg Leu Tyr Arg Asn Phe Glu Arg Phe Arg Arg
245 250 255
Ala Glu Leu Leu Asp Ala Ala Lys Ala Gly Gly Glu Phe Leu Leu Arg
260 265 270
Phe Ala Arg Val Ala Pro Pro Ala Lys Lys Cys Ala Phe Val Leu Thr
275 280 285
Arg Asp Gly Arg Pro Val Lys Val Gln Arg Thr Ile Phe Ser Glu Cys
290 295 300
Phe Tyr Thr Ile Ala Met Asn Glu Leu Trp Arg Val Thr Lys Glu Ser
305 310 315 320
Arg Tyr Gln Asn Glu Ala Val Glu Met Met Asp Gln Ile Val His Trp
325 330 335
Val Arg Glu Asp Pro Ala Gly Leu Gly Arg Pro Gln Leu Ser Gly Thr
340 345 350
Pro Asp Ala Glu Ser Met Ala Val Pro Met Met Leu Met Asn Leu Val
355 360 365
Asp Gln Leu Ser Glu Glu Asp Glu Ala Leu Thr Lys Lys Tyr Ala Glu
370 375 380
Leu Trp Asp Trp Cys Ala Gln Arg Ile Leu Gln His Val Gln Arg Asp
385 390 395 400
Gly Lys Ala Ile Leu Glu Asn Val Ser His Asp Gly Lys Glu Leu Thr
405 410 415
Gly Cys Leu Gly Arg His Gln Asn Pro Gly His Ala Ile Glu Ala Gly
420 425 430
Trp Phe Leu Leu Gln Tyr Ala Arg Lys Lys Gly Asp Ala Lys Leu Arg
435 440 445
Ala His Ile Ile Asp Lys Phe Leu Leu Leu Pro Phe Arg Ser Gly Trp
450 455 460
Asp Ala Glu His Gly Gly Leu Phe Tyr Phe Gln Asp Val Asp Gly Phe
465 470 475 480
Cys Pro Thr Gln Leu Glu Trp Asp Met Lys Leu Trp Trp Pro His Asn
485 490 495
Glu Ala Met Ile Ser Phe Leu Met Gly Tyr Arg Asp Ser Gly Asp Pro
500 505 510
Ala Leu Leu Gln Ile Phe Asn Gln Val Ala Glu Tyr Thr Phe Lys His
515 520 525
Phe His Asp Pro Glu Asn Gly Glu Trp Phe Gly Tyr Leu Asn Arg Glu
530 535 540
Gly Lys Val Ala Leu Asn Phe Lys Gly Gly Pro Phe Lys Gly Cys Phe
545 550 555 560
His Val Pro Arg Cys Leu Ala Met Cys Glu Lys Met Leu Gly Asp Leu
565 570 575
Leu His Arg Gly Ser Ala Pro Ala Gly Ser Lys
580 585
<210> 83
<211> 1818
<212> ДНК
<213> Homo sapiens
<220>
<221> misc_feature
<223> hWip1
<400> 83
atggcggggc tgtactcgct gggagtgagc gtcttctccg accagggcgg gaggaagtac 60
atggaggacg ttactcaaat cgttgtggag cccgaaccga cggctgaaga aaagccctcg 120
ccgcggcggt cgctgtctca gccgttgcct ccgcggccgt cgccggccgc ccttcccggc 180
ggcgaagtct cggggaaagg cccagcggtg gcagcccgag aggctcgcga ccctctcccg 240
gacgccgggg cctcgccggc acctagccgc tgctgccgcc gccgttcctc cgtggccttt 300
ttcgccgtgt gcgacgggca cggcgggcgg gaggcggcac agtttgcccg ggagcacttg 360
tggggtttca tcaagaagca gaagggtttc acctcgtccg agccggctaa ggtttgcgct 420
gccatccgca aaggctttct cgcttgtcac cttgccatgt ggaagaaact ggcggaatgg 480
ccaaagacta tgacgggtct tcctagcaca tcagggacaa ctgccagtgt ggtcatcatt 540
cggggcatga agatgtatgt agctcacgta ggtgactcag gggtggttct tggaattcag 600
gatgacccga aggatgactt tgtcagagct gtggaggtga cacaggacca taagccagaa 660
cttcccaagg aaagagaacg aatcgaagga cttggtggga gtgtaatgaa caagtctggg 720
gtgaatcgtg tagtttggaa acgacctcga ctcactcaca atggacctgt tagaaggagc 780
acagttattg accagattcc ttttctggca gtagcaagag cacttggtga tttgtggagc 840
tatgatttct tcagtggtga atttgtggtg tcacctgaac cagacacaag tgtccacact 900
cttgaccctc agaagcacaa gtatattata ttggggagtg atggactttg gaatatgatt 960
ccaccacaag atgccatctc aatgtgccag gaccaagagg agaaaaaata cctgatgggt 1020
gagcatggac aatcttgtgc caaaatgctt gtgaatcgag cattgggccg ctggaggcag 1080
cgtatgctcc gagcagataa cactagtgcc atagtaatct gcatctctcc agaagtggac 1140
aatcagggaa actttaccaa tgaagatgag ttatacctga acctgactga cagcccttcc 1200
tataatagtc aagaaacctg tgtgatgact ccttccccat gttctacacc accagtcaag 1260
tcactggagg aggatccatg gccaagggtg aattctaagg accatatacc tgccctggtt 1320
cgtagcaatg ccttctcaga gaatttttta gaggtttcag ctgagatagc tcgagagaat 1380
gtccaaggtg tagtcatacc ctcaaaagat ccagaaccac ttgaagaaaa ttgcgctaaa 1440
gccctgactt taaggataca tgattctttg aataatagcc ttccaattgg ccttgtgcct 1500
actaattcaa caaacactgt catggaccaa aaaaatttga agatgtcaac tcctggccaa 1560
atgaaagccc aagaaattga aagaacccct ccaacaaact ttaaaaggac attagaagag 1620
tccaattctg gccccctgat gaagaagcat agacgaaatg gcttaagtcg aagtagtggt 1680
gctcagcctg caagtctccc cacaacctca cagcgaaaga actctgttaa actcaccatg 1740
cgacgcagac ttaggggcca gaagaaaatt ggaaatcctt tacttcatca acacaggaaa 1800
actgtttgtg tttgctga 1818
<210> 84
<211> 605
<212> ПРТ
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<223> hWip1
<400> 84
Met Ala Gly Leu Tyr Ser Leu Gly Val Ser Val Phe Ser Asp Gln Gly
1 5 10 15
Gly Arg Lys Tyr Met Glu Asp Val Thr Gln Ile Val Val Glu Pro Glu
20 25 30
Pro Thr Ala Glu Glu Lys Pro Ser Pro Arg Arg Ser Leu Ser Gln Pro
35 40 45
Leu Pro Pro Arg Pro Ser Pro Ala Ala Leu Pro Gly Gly Glu Val Ser
50 55 60
Gly Lys Gly Pro Ala Val Ala Ala Arg Glu Ala Arg Asp Pro Leu Pro
65 70 75 80
Asp Ala Gly Ala Ser Pro Ala Pro Ser Arg Cys Cys Arg Arg Arg Ser
85 90 95
Ser Val Ala Phe Phe Ala Val Cys Asp Gly His Gly Gly Arg Glu Ala
100 105 110
Ala Gln Phe Ala Arg Glu His Leu Trp Gly Phe Ile Lys Lys Gln Lys
115 120 125
Gly Phe Thr Ser Ser Glu Pro Ala Lys Val Cys Ala Ala Ile Arg Lys
130 135 140
Gly Phe Leu Ala Cys His Leu Ala Met Trp Lys Lys Leu Ala Glu Trp
145 150 155 160
Pro Lys Thr Met Thr Gly Leu Pro Ser Thr Ser Gly Thr Thr Ala Ser
165 170 175
Val Val Ile Ile Arg Gly Met Lys Met Tyr Val Ala His Val Gly Asp
180 185 190
Ser Gly Val Val Leu Gly Ile Gln Asp Asp Pro Lys Asp Asp Phe Val
195 200 205
Arg Ala Val Glu Val Thr Gln Asp His Lys Pro Glu Leu Pro Lys Glu
210 215 220
Arg Glu Arg Ile Glu Gly Leu Gly Gly Ser Val Met Asn Lys Ser Gly
225 230 235 240
Val Asn Arg Val Val Trp Lys Arg Pro Arg Leu Thr His Asn Gly Pro
245 250 255
Val Arg Arg Ser Thr Val Ile Asp Gln Ile Pro Phe Leu Ala Val Ala
260 265 270
Arg Ala Leu Gly Asp Leu Trp Ser Tyr Asp Phe Phe Ser Gly Glu Phe
275 280 285
Val Val Ser Pro Glu Pro Asp Thr Ser Val His Thr Leu Asp Pro Gln
290 295 300
Lys His Lys Tyr Ile Ile Leu Gly Ser Asp Gly Leu Trp Asn Met Ile
305 310 315 320
Pro Pro Gln Asp Ala Ile Ser Met Cys Gln Asp Gln Glu Glu Lys Lys
325 330 335
Tyr Leu Met Gly Glu His Gly Gln Ser Cys Ala Lys Met Leu Val Asn
340 345 350
Arg Ala Leu Gly Arg Trp Arg Gln Arg Met Leu Arg Ala Asp Asn Thr
355 360 365
Ser Ala Ile Val Ile Cys Ile Ser Pro Glu Val Asp Asn Gln Gly Asn
370 375 380
Phe Thr Asn Glu Asp Glu Leu Tyr Leu Asn Leu Thr Asp Ser Pro Ser
385 390 395 400
Tyr Asn Ser Gln Glu Thr Cys Val Met Thr Pro Ser Pro Cys Ser Thr
405 410 415
Pro Pro Val Lys Ser Leu Glu Glu Asp Pro Trp Pro Arg Val Asn Ser
420 425 430
Lys Asp His Ile Pro Ala Leu Val Arg Ser Asn Ala Phe Ser Glu Asn
435 440 445
Phe Leu Glu Val Ser Ala Glu Ile Ala Arg Glu Asn Val Gln Gly Val
450 455 460
Val Ile Pro Ser Lys Asp Pro Glu Pro Leu Glu Glu Asn Cys Ala Lys
465 470 475 480
Ala Leu Thr Leu Arg Ile His Asp Ser Leu Asn Asn Ser Leu Pro Ile
485 490 495
Gly Leu Val Pro Thr Asn Ser Thr Asn Thr Val Met Asp Gln Lys Asn
500 505 510
Leu Lys Met Ser Thr Pro Gly Gln Met Lys Ala Gln Glu Ile Glu Arg
515 520 525
Thr Pro Pro Thr Asn Phe Lys Arg Thr Leu Glu Glu Ser Asn Ser Gly
530 535 540
Pro Leu Met Lys Lys His Arg Arg Asn Gly Leu Ser Arg Ser Ser Gly
545 550 555 560
Ala Gln Pro Ala Ser Leu Pro Thr Thr Ser Gln Arg Lys Asn Ser Val
565 570 575
Lys Leu Thr Met Arg Arg Arg Leu Arg Gly Gln Lys Lys Ile Gly Asn
580 585 590
Pro Leu Leu His Gln His Arg Lys Thr Val Cys Val Cys
595 600 605
<210> 85
<211> 741
<212> ДНК
<213> Homo sapiens
<220>
<221> misc_feature
<223> hRTP4
<400> 85
atggttgtag atttctggac ttgggagcag acatttcaag aactaatcca agaggcaaaa 60
ccccgggcca catggacgct gaagttggat ggcaaccttc agctagactg cctggctcaa 120
gggtggaagc aataccaaca gagagcattt ggctggttcc ggtgttcctc ctgccagcga 180
agttgggctt ccgcccaagt gcagattctg tgccacacgt actgggagca ctggacatcc 240
cagggtcagg tgcgtatgag gctctttggc caaaggtgcc agaagtgctc ctggtcccaa 300
tatgagatgc ctgagttctc ctcggatagc accatgagga ttctgagcaa cctggtgcag 360
catatactga agaaatacta tggaaatggc acgaggaagt ctccagaaat gccagtaatc 420
ctggaagtgt ccctggaagg atcccatgac acagccaatt gtgaggcatg cactttgggc 480
atctgtggac agggcttaaa aagctgcatg acaaagccgt ccaaatccct actcccccac 540
ctaaagactg ggaattcctc acctggaatt ggtgctgtgt acctcgcaaa ccaagccaag 600
aaccagtcag ctgaggcaaa agaggctaag gggagtgggt atgagaaatt agggcccagt 660
cgagacccag atccactgaa catctgtgtc tttattttgc tgcttgtatt tattgtagtc 720
aaatgcttta catcagaatg a 741
<210> 86
<211> 246
<212> ПРТ
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<223> hRTP4
<400> 86
Met Val Val Asp Phe Trp Thr Trp Glu Gln Thr Phe Gln Glu Leu Ile
1 5 10 15
Gln Glu Ala Lys Pro Arg Ala Thr Trp Thr Leu Lys Leu Asp Gly Asn
20 25 30
Leu Gln Leu Asp Cys Leu Ala Gln Gly Trp Lys Gln Tyr Gln Gln Arg
35 40 45
Ala Phe Gly Trp Phe Arg Cys Ser Ser Cys Gln Arg Ser Trp Ala Ser
50 55 60
Ala Gln Val Gln Ile Leu Cys His Thr Tyr Trp Glu His Trp Thr Ser
65 70 75 80
Gln Gly Gln Val Arg Met Arg Leu Phe Gly Gln Arg Cys Gln Lys Cys
85 90 95
Ser Trp Ser Gln Tyr Glu Met Pro Glu Phe Ser Ser Asp Ser Thr Met
100 105 110
Arg Ile Leu Ser Asn Leu Val Gln His Ile Leu Lys Lys Tyr Tyr Gly
115 120 125
Asn Gly Thr Arg Lys Ser Pro Glu Met Pro Val Ile Leu Glu Val Ser
130 135 140
Leu Glu Gly Ser His Asp Thr Ala Asn Cys Glu Ala Cys Thr Leu Gly
145 150 155 160
Ile Cys Gly Gln Gly Leu Lys Ser Cys Met Thr Lys Pro Ser Lys Ser
165 170 175
Leu Leu Pro His Leu Lys Thr Gly Asn Ser Ser Pro Gly Ile Gly Ala
180 185 190
Val Tyr Leu Ala Asn Gln Ala Lys Asn Gln Ser Ala Glu Ala Lys Glu
195 200 205
Ala Lys Gly Ser Gly Tyr Glu Lys Leu Gly Pro Ser Arg Asp Pro Asp
210 215 220
Pro Leu Asn Ile Cys Val Phe Ile Leu Leu Leu Val Phe Ile Val Val
225 230 235 240
Lys Cys Phe Thr Ser Glu
245
<210> 87
<211> 765
<212> ДНК
<213> Homo sapiens
<220>
<221> misc_feature
<223> hREEP2
<400> 87
atggtgtcct ggatcatctc tcgcctggtg gtgctcatct ttggcaccct gtacccagcc 60
tattcttcct acaaggccgt gaagacaaaa aacgtgaagg aatatgtgaa atggatgatg 120
tactggatcg tctttgcctt cttcaccacg gccgagacgc tcacggatat agtgctctcc 180
tggttcccct tctactttga actgaagatc gccttcgtga tatggctgct gtccccttac 240
accaagggct ccagcgtgct ctaccgcaag ttcgtgcacc caacgctgtc caacaaggag 300
aaggagatcg acgagtacat cacgcaggcc cgagacaaga gctatgagac catgatgagg 360
gtgggcaaga ggggcctgaa ccttgccgcc aatgctgcag tcacagctgc cgccaagggc 420
cagggggtgc tgtcagagaa gctccgcagc ttcagcatgc aggacctgac cctgatccgg 480
gacgaggacg cactgcccct gcagaggcct gacggccgcc tccgacccag ccctggcagc 540
ctcctggaca ccatcgagga cttaggagat gaccctgccc tgagtctaag gtccagcaca 600
aacccggcag attcccggac agaggcttct gaggatgaca tgggagacaa agctcccaag 660
agggccaaac ccatcaaaaa agcgcccaaa gctgagccac tggcttccaa gacactgaag 720
acccggccca agaagaagac ctctggcggg ggcgactcag cttga 765
<210> 88
<211> 254
<212> ПРТ
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<223> hREEP2
<400> 88
Met Val Ser Trp Ile Ile Ser Arg Leu Val Val Leu Ile Phe Gly Thr
1 5 10 15
Leu Tyr Pro Ala Tyr Ser Ser Tyr Lys Ala Val Lys Thr Lys Asn Val
20 25 30
Lys Glu Tyr Val Lys Trp Met Met Tyr Trp Ile Val Phe Ala Phe Phe
35 40 45
Thr Thr Ala Glu Thr Leu Thr Asp Ile Val Leu Ser Trp Phe Pro Phe
50 55 60
Tyr Phe Glu Leu Lys Ile Ala Phe Val Ile Trp Leu Leu Ser Pro Tyr
65 70 75 80
Thr Lys Gly Ser Ser Val Leu Tyr Arg Lys Phe Val His Pro Thr Leu
85 90 95
Ser Asn Lys Glu Lys Glu Ile Asp Glu Tyr Ile Thr Gln Ala Arg Asp
100 105 110
Lys Ser Tyr Glu Thr Met Met Arg Val Gly Lys Arg Gly Leu Asn Leu
115 120 125
Ala Ala Asn Ala Ala Val Thr Ala Ala Ala Lys Gly Gln Gly Val Leu
130 135 140
Ser Glu Lys Leu Arg Ser Phe Ser Met Gln Asp Leu Thr Leu Ile Arg
145 150 155 160
Asp Glu Asp Ala Leu Pro Leu Gln Arg Pro Asp Gly Arg Leu Arg Pro
165 170 175
Ser Pro Gly Ser Leu Leu Asp Thr Ile Glu Asp Leu Gly Asp Asp Pro
180 185 190
Ala Leu Ser Leu Arg Ser Ser Thr Asn Pro Ala Asp Ser Arg Thr Glu
195 200 205
Ala Ser Glu Asp Asp Met Gly Asp Lys Ala Pro Lys Arg Ala Lys Pro
210 215 220
Ile Lys Lys Ala Pro Lys Ala Glu Pro Leu Ala Ser Lys Thr Leu Lys
225 230 235 240
Thr Arg Pro Lys Lys Lys Thr Ser Gly Gly Gly Asp Ser Ala
245 250
<210> 89
<211> 783
<212> ДНК
<213> Homo sapiens
<220>
<221> misc_feature
<223> hDPM1
<400> 89
atggcctcct tggaagtcag tcgtagtcct cgcaggtctc ggcgggagct ggaagtgcgc 60
agtccacgac agaacaaata ttcggtgctt ttacctacct acaacgagcg cgagaacctg 120
ccgctcatcg tgtggctgct ggtgaaaagc ttctccgaga gtggaatcaa ctatgaaatt 180
ataatcatag atgatggaag cccagatgga acaagggatg ttgctgaaca gttggagaag 240
atctatgggt cagacagaat tcttctaaga ccacgagaga aaaagttggg actaggaact 300
gcatatattc atggaatgaa acatgccaca ggaaactaca tcattattat ggatgctgat 360
ctctcacacc atccaaaatt tattcctgaa tttattagga agcaaaagga gggtaatttt 420
gatattgtct ctggaactcg ctacaaagga aatggaggtg tatatggctg ggatttgaaa 480
agaaaaataa tcagccgtgg ggccaatttt ttaactcaga tcttgctgag accaggagca 540
tctgatttaa caggaagttt cagattatac cgaaaagaag ttctagagaa attaatagaa 600
aaatgtgttt ctaaaggcta cgtcttccag atggagatga ttgttcgggc aagacagttg 660
aattatacta ttggcgaggt tccaatatca tttgtggatc gtgtttatgg tgaatccaag 720
ttgggaggaa atgaaatagt atctttcttg aaaggattat tgactctttt tgctactaca 780
taa 783
<210> 90
<211> 260
<212> ПРТ
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<223> hDPM1
<400> 90
Met Ala Ser Leu Glu Val Ser Arg Ser Pro Arg Arg Ser Arg Arg Glu
1 5 10 15
Leu Glu Val Arg Ser Pro Arg Gln Asn Lys Tyr Ser Val Leu Leu Pro
20 25 30
Thr Tyr Asn Glu Arg Glu Asn Leu Pro Leu Ile Val Trp Leu Leu Val
35 40 45
Lys Ser Phe Ser Glu Ser Gly Ile Asn Tyr Glu Ile Ile Ile Ile Asp
50 55 60
Asp Gly Ser Pro Asp Gly Thr Arg Asp Val Ala Glu Gln Leu Glu Lys
65 70 75 80
Ile Tyr Gly Ser Asp Arg Ile Leu Leu Arg Pro Arg Glu Lys Lys Leu
85 90 95
Gly Leu Gly Thr Ala Tyr Ile His Gly Met Lys His Ala Thr Gly Asn
100 105 110
Tyr Ile Ile Ile Met Asp Ala Asp Leu Ser His His Pro Lys Phe Ile
115 120 125
Pro Glu Phe Ile Arg Lys Gln Lys Glu Gly Asn Phe Asp Ile Val Ser
130 135 140
Gly Thr Arg Tyr Lys Gly Asn Gly Gly Val Tyr Gly Trp Asp Leu Lys
145 150 155 160
Arg Lys Ile Ile Ser Arg Gly Ala Asn Phe Leu Thr Gln Ile Leu Leu
165 170 175
Arg Pro Gly Ala Ser Asp Leu Thr Gly Ser Phe Arg Leu Tyr Arg Lys
180 185 190
Glu Val Leu Glu Lys Leu Ile Glu Lys Cys Val Ser Lys Gly Tyr Val
195 200 205
Phe Gln Met Glu Met Ile Val Arg Ala Arg Gln Leu Asn Tyr Thr Ile
210 215 220
Gly Glu Val Pro Ile Ser Phe Val Asp Arg Val Tyr Gly Glu Ser Lys
225 230 235 240
Leu Gly Gly Asn Glu Ile Val Ser Phe Leu Lys Gly Leu Leu Thr Leu
245 250 255
Phe Ala Thr Thr
260
<210> 91
<211> 2109
<212> ДНК
<213> Homo sapiens
<220>
<221> misc_feature
<223> hDRiP78
<400> 91
atggcccaga agcaccccgg agaaagaggg ttgtatggag cccaccacag tggtggtgcc 60
tccctcagga ctttaggacc ctccgtggac cctgaaatac cttcattctc aggactcagg 120
gactcagcag ggactgctcc taatggtacc cgctgcctca cagagcactc tggtcctaag 180
cacacacagc acccaaaccc agcccattgg ttggacccaa gccatggccc cccagggggt 240
ccaggaccac ctagagatgc agaggaccct gatcagagtg agacgtcttc agaagaagaa 300
tcaggagtgg accaggaact ctcaaaagaa aacgagactg ggaaccagaa ggatgggaac 360
tcttttcttt ccattccatc tgcttgcaac tgccagggaa cacctggaat tccagaaggg 420
ccttactctg agggaggaaa tggttcttct agcaactttt gccaccactg tacctctcca 480
gctttggggg aagatgagtt ggaagaggaa tatgatgatg aagaatctct caagttcccc 540
agtgattttt cacgtgtgtc cagcggaaag aaacccccat cccggagaca gcggcaccgc 600
tttccaacga aggaggatac tcgggagggt ggacgtaggg atcccaggtc ccctggtcga 660
catcggctgg gtcggaaacg aagtcaggca gataagcgca aaggcctggg attgtgggga 720
gccgaggaac tatgtcaact tggacaggca ggcttttggt ggctgattga actgctggta 780
ttggtgggag agtacgtaga aacttgtggc catctcatct atgcctgcag gcaactgaaa 840
agcagtgatt tggacctttt tcgagtttgg atgggagtgt ggacagggcg gttagggggc 900
tgggcccagg tcatgtttca gtttctaagc caggggtttt actgtggagt aggactgttt 960
actcgttttc ttaagctgct gggtgctttg ctgctcctgg ctctggccct ctttttgggc 1020
tttctacagt tgggatggcg gtttctggtg ggactaggtg accggttagg ctggagggat 1080
aaggctacct ggctcttctc ttggctggat tctccagcct tgcagcgttg cttgactctg 1140
ctgagagata gcaggccatg gcagcggctg gtaagaatag ttcagtgggg ctggctggag 1200
ttgccttggg tcaagcagaa tattaatagg caggggaatg cacctgtagc tagtgggcgc 1260
tactgccagc ctgaagagga agtggctcga ctcttgacca tggctggggt tcctgaggat 1320
gagctaaacc ctttccatgt actgggggtt gaggccacag catcagatgt tgaactgaag 1380
aaggcctata gacagctggc agtgatggtt catcctgaca aaaatcatca tccccgggct 1440
gaggaggcct tcaaggtttt gcgagcagct tgggacattg tcagcaatgc tgaaaagcga 1500
aaggagtatg agatgaaacg aatggcagag aatgagctga gccggtcagt aaatgagttt 1560
ctgtccaagc tgcaagatga cctcaaggag gcaatgaata ctatgatgtg tagccgatgc 1620
caaggaaagc ataggaggtt tgaaatggac cgggaaccta agagtgccag atactgtgct 1680
gagtgtaata ggctgcatcc tgctgaggaa ggagactttt gggcagagtc aagcatgttg 1740
ggcctcaaga tcacctactt tgcactgatg gatggaaagg tgtatgacat cacagagtgg 1800
gctggatgcc agcgtgtagg tatctcccca gatacccaca gagtccccta tcacatctca 1860
tttggttctc ggattccagg caccagaggg cggcagagag ccaccccaga tgcccctcct 1920
gctgatcttc aggatttctt gagtcggatc tttcaagtac ccccagggca gatgcccaat 1980
gggaacttct ttgcagctcc tcagcctgcc cctggagccg ctgcagcctc taagcccaac 2040
agcacagtac ccaagggaga agccaaacct aagcggcgga agaaagtgag gaggcccttc 2100
caacgttga 2109
<210> 92
<211> 702
<212> ПРТ
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<223> hDRiP78
<400> 92
Met Ala Gln Lys His Pro Gly Glu Arg Gly Leu Tyr Gly Ala His His
1 5 10 15
Ser Gly Gly Ala Ser Leu Arg Thr Leu Gly Pro Ser Val Asp Pro Glu
20 25 30
Ile Pro Ser Phe Ser Gly Leu Arg Asp Ser Ala Gly Thr Ala Pro Asn
35 40 45
Gly Thr Arg Cys Leu Thr Glu His Ser Gly Pro Lys His Thr Gln His
50 55 60
Pro Asn Pro Ala His Trp Leu Asp Pro Ser His Gly Pro Pro Gly Gly
65 70 75 80
Pro Gly Pro Pro Arg Asp Ala Glu Asp Pro Asp Gln Ser Glu Thr Ser
85 90 95
Ser Glu Glu Glu Ser Gly Val Asp Gln Glu Leu Ser Lys Glu Asn Glu
100 105 110
Thr Gly Asn Gln Lys Asp Gly Asn Ser Phe Leu Ser Ile Pro Ser Ala
115 120 125
Cys Asn Cys Gln Gly Thr Pro Gly Ile Pro Glu Gly Pro Tyr Ser Glu
130 135 140
Gly Gly Asn Gly Ser Ser Ser Asn Phe Cys His His Cys Thr Ser Pro
145 150 155 160
Ala Leu Gly Glu Asp Glu Leu Glu Glu Glu Tyr Asp Asp Glu Glu Ser
165 170 175
Leu Lys Phe Pro Ser Asp Phe Ser Arg Val Ser Ser Gly Lys Lys Pro
180 185 190
Pro Ser Arg Arg Gln Arg His Arg Phe Pro Thr Lys Glu Asp Thr Arg
195 200 205
Glu Gly Gly Arg Arg Asp Pro Arg Ser Pro Gly Arg His Arg Leu Gly
210 215 220
Arg Lys Arg Ser Gln Ala Asp Lys Arg Lys Gly Leu Gly Leu Trp Gly
225 230 235 240
Ala Glu Glu Leu Cys Gln Leu Gly Gln Ala Gly Phe Trp Trp Leu Ile
245 250 255
Glu Leu Leu Val Leu Val Gly Glu Tyr Val Glu Thr Cys Gly His Leu
260 265 270
Ile Tyr Ala Cys Arg Gln Leu Lys Ser Ser Asp Leu Asp Leu Phe Arg
275 280 285
Val Trp Met Gly Val Trp Thr Gly Arg Leu Gly Gly Trp Ala Gln Val
290 295 300
Met Phe Gln Phe Leu Ser Gln Gly Phe Tyr Cys Gly Val Gly Leu Phe
305 310 315 320
Thr Arg Phe Leu Lys Leu Leu Gly Ala Leu Leu Leu Leu Ala Leu Ala
325 330 335
Leu Phe Leu Gly Phe Leu Gln Leu Gly Trp Arg Phe Leu Val Gly Leu
340 345 350
Gly Asp Arg Leu Gly Trp Arg Asp Lys Ala Thr Trp Leu Phe Ser Trp
355 360 365
Leu Asp Ser Pro Ala Leu Gln Arg Cys Leu Thr Leu Leu Arg Asp Ser
370 375 380
Arg Pro Trp Gln Arg Leu Val Arg Ile Val Gln Trp Gly Trp Leu Glu
385 390 395 400
Leu Pro Trp Val Lys Gln Asn Ile Asn Arg Gln Gly Asn Ala Pro Val
405 410 415
Ala Ser Gly Arg Tyr Cys Gln Pro Glu Glu Glu Val Ala Arg Leu Leu
420 425 430
Thr Met Ala Gly Val Pro Glu Asp Glu Leu Asn Pro Phe His Val Leu
435 440 445
Gly Val Glu Ala Thr Ala Ser Asp Val Glu Leu Lys Lys Ala Tyr Arg
450 455 460
Gln Leu Ala Val Met Val His Pro Asp Lys Asn His His Pro Arg Ala
465 470 475 480
Glu Glu Ala Phe Lys Val Leu Arg Ala Ala Trp Asp Ile Val Ser Asn
485 490 495
Ala Glu Lys Arg Lys Glu Tyr Glu Met Lys Arg Met Ala Glu Asn Glu
500 505 510
Leu Ser Arg Ser Val Asn Glu Phe Leu Ser Lys Leu Gln Asp Asp Leu
515 520 525
Lys Glu Ala Met Asn Thr Met Met Cys Ser Arg Cys Gln Gly Lys His
530 535 540
Arg Arg Phe Glu Met Asp Arg Glu Pro Lys Ser Ala Arg Tyr Cys Ala
545 550 555 560
Glu Cys Asn Arg Leu His Pro Ala Glu Glu Gly Asp Phe Trp Ala Glu
565 570 575
Ser Ser Met Leu Gly Leu Lys Ile Thr Tyr Phe Ala Leu Met Asp Gly
580 585 590
Lys Val Tyr Asp Ile Thr Glu Trp Ala Gly Cys Gln Arg Val Gly Ile
595 600 605
Ser Pro Asp Thr His Arg Val Pro Tyr His Ile Ser Phe Gly Ser Arg
610 615 620
Ile Pro Gly Thr Arg Gly Arg Gln Arg Ala Thr Pro Asp Ala Pro Pro
625 630 635 640
Ala Asp Leu Gln Asp Phe Leu Ser Arg Ile Phe Gln Val Pro Pro Gly
645 650 655
Gln Met Pro Asn Gly Asn Phe Phe Ala Ala Pro Gln Pro Ala Pro Gly
660 665 670
Ala Ala Ala Ala Ser Lys Pro Asn Ser Thr Val Pro Lys Gly Glu Ala
675 680 685
Lys Pro Lys Arg Arg Lys Lys Val Arg Arg Pro Phe Gln Arg
690 695 700
<210> 93
<211> 1020
<212> ДНК
<213> Cricetulus griseus
<220>
<221> misc_feature
<223> Rad51
<400> 93
atggctatgc agatgcagct tgaagcaaat gcagatactt cagtggaaga agaaagcttt 60
ggtccacaac ctatttcacg gttagagcaa tgtggcataa gtgccaatga tgtgaagaaa 120
ttagaagaag ctggtttcca tacggtggag gctgttgctt atgcaccaaa gaaggaactc 180
ataaatatta agggaattag tgaagccaaa gcagacaaaa ttctggctga ggccgctaaa 240
ttagttccaa tgggtttcac cactgcaact gaatttcacc aaaggcgttc agaaatcata 300
cagattacta ctggctccaa agagcttgac aaactgcttc aaggtggaat tgagactgga 360
tctatcacag agatgtttgg agaattccga actgggaaga cacagatctg tcatacattg 420
gctgtaacat gccagcttcc cattgatcgt ggtggaggtg aaggaaaggc catgtacatt 480
gacaccgagg gtacgtttag gccagaacgg ctgctagcag tggctgagag gtatggtctg 540
tctggcagcg atgtcctaga taatgtagca tatgctcgag ggttcaacac agaccaccaa 600
acccagctcc tttatcaagc atcagccatg atggtagaat ccagatatgc actgcttatt 660
gtagacagtg ctactgccct ctacagaaca gactactcag gtcgaggaga gctttcagcc 720
aggcaaatgc atttggccag atttctgagg atgctgctgc gacttgctga tgagtttggt 780
gtagcagtgg taatcaccaa ccaggtagta gcccaagtgg atggagcagc catgttcact 840
gcagatccca aaaaacccat tggaggaaac atcattgccc atgcatcaac aaccaggctg 900
tacctgagga aaggaagagg ggagaccaga atctgcaaag tctatgactc tccctgtctc 960
cctgaagctg aagccatgtt tgccattaat gcagatggag taggagatgc caaggactga 1020
<210> 94
<211> 1053
<212> ДНК
<213> Cricetulus griseus
<220>
<221> misc_feature
<223> Rad51b
<400> 94
atgagcaaca agaaactaag acgagtaggt ttatcacaag agctgtgtga ccgtttgagc 60
agacatcaga ttgttaattg tcaggacttt ttaggtcttt ccccactgga acttatgaaa 120
gtgactggcc tgagttatgg aggtgtccag gagcttctgt atatggtcag cagggcctgt 180
gccccacaga tgcaaacagc ctatgatata aagatgcgga ggtctgctga actctcccca 240
gcgttcctgt ctactaccct ttctgctttg gacaaagccc tgcatggtgg tgtggcttgt 300
ggatcgctca cagagattac aggtccgcca ggttgtggga aaactcagtt ttgtataatg 360
atgagtgttt tagctacatt gcccaccaac atgggaggat tagaagggac tgtcgtgtat 420
attgacaccg agtctgcgtt tactgctgaa agactggtcg agattgcaga atcccgtttt 480
ccactctatt ttaacacaga agaaaaactg cttttgatga gcagtaaagt tcatcttcac 540
cgggaactca gctgtgaggc agttctgcaa aggcttgaat ctttggagga agagattatt 600
tccaaaggag ttaagcttgt gattgttgac tctattgctt ctgtggtcag aaaggagttc 660
gatcctcagc ttcagggcaa catcaaagaa aggaacaagt tcttggccaa acaagcatcc 720
ttgctgaagt acctggctga ggaattttca ctcccagtta tcttgacgaa tcaaattacg 780
acccatctga gtggagccct cccttctcaa gcagacctgg tgtctccggc tgatgatttg 840
tccctgtctg aaggcacttc tggatccagc tgtgtggtag ccgcactggg aaactcatgg 900
agtcactgtg tgaacacccg gctgattctc cagtaccttg actcagagag aaggcagatt 960
ctcattgcca agtcacctct ggctgccttc acctccttca tctataccat caagggggaa 1020
ggcctagttc ttcaaggcca agaaaggcca tag 1053
<210> 95
<211> 1101
<212> ДНК
<213> Cricetulus griseus
<220>
<221> misc_feature
<223> Rad51c
<400> 95
atgcagcggg agctagtgag tttcccgctg tctcccacgg tgcgagtgaa gctggtggct 60
gcgggtttcc agacggccga ggacgtcctg ggggtgaagc cctctgagct cagcaaagaa 120
gttgggatat ctaaagagga agctttagaa actctacaaa ttgtaagaag agagagtctc 180
acagacaaac ccagatgtgc tggtgcatct gtggcaggca agaagtacac cgcactggaa 240
cttcttgagc aggagcacac ccagggcttc ataatcacct tctgctcagc gctagataac 300
attcttgggg gtggaatacc cctaatgaaa acaacggaag tttgtggtgt accaggtgtt 360
ggaaaaacac agttatgtat gcagttggca gtggatgtgc agattccaga gtgttttgga 420
ggagtggcag gtgaagcagt gtttattgat acggagggaa gttttatggt tgatagagtg 480
gtcacccttg caaatgcctg cattcagcac cttcacctta tagcaggaac acacaaggat 540
gaagaacacc agaaagcctt ggagggcttt actcttgaaa atattctttc tcatatttat 600
tatttccgtt gtcatgatta tactgagttg ctggcacaag tctatctcct tccagatttc 660
ctttcaaatc attcaaaggt gcagttagtg ataatagatg gcattgctct tccttttcga 720
catgaccttg atgatctatc ccttcgtact cgattactaa atggccttgc ccaacaaatg 780
atcagcctgg caaataatca cagattagct gttattttaa ctaatcagat gacaacaaag 840
attgataaaa atcaagcatt gcttgttcct gcattagggg aaagctgggg acatgctgct 900
acaataagac ttatttttca ctgggagcaa aagcaaaggt ttgcaacatt gtacaagtca 960
ccaagccaga aggagtccac aataccattt cagatcacac cacagggatt tagagatgct 1020
gctgtcactg cttcttcatc acagacagaa ggttcttcaa atctccggaa acggtcacga 1080
gaaccagagg aaggatgctg a 1101
<210> 96
<211> 1002
<212> ДНК
<213> Cricetulus griseus
<220>
<221> misc_feature
<223> Rad51d
<400> 96
aaacatgggc gtgctcaggg tggggctgtg cccgggcctc accgaagaga tcgtccagct 60
tctgaagggc caaaggatca agacagtggc ggacctggca gctgctaacc tggaggaggt 120
agcccagaag tgtggcttgt cctacaaggc cctggttgcc ctgaggaggg tattgctggc 180
gcagttctcg gctttcccat taaacggagc agacctatat gaggaactga agacttccac 240
tgccatcctg tccacaggca ttggaagcct ggacaaacta cttgatgctg gcctctatac 300
aggggaggtg actgaaattg taggaggtcc aggtagcgga aaaacccagg tgtgcctgtg 360
tgtggctgca aatgtggccc atagcctgca gcagaatgta ctgtatgttg attccagtgg 420
aggaatgaca gcatcccgcc tcctgcagct actacaggct agaacccaag atgaggagaa 480
acaggcaggt gctctccaga ggatacaggt ggtgcatgta tttgacatct tccagatgct 540
ggatatgcta caggaccttc ggggctccat ggcccagcag tcgacatctt cttcaggcac 600
tgtgaaggtt gtgattgtgg attctgtcac tgccgtgatt gccccacttc tgggaggtca 660
gcagagggaa ggcctggcct tgatgatgca gctggcccga gagctcaaga tcctggcccg 720
ggacctagct gtggcagtgg tggtgaccaa ccacttgacc cgagacaggg atggtaggag 780
gttcaaacct gctctgggac gctcctggag ctttgtgccc agtacccgga ttctcctgga 840
tgtcaccaaa ggggctggaa cattaggcag aggccaacgc acagtgtgtc tgaccaagtc 900
tccccgccag ccaacaggtc tacaggaggt gatagacatt gggacattgg ggactgagga 960
gcagagccca gaattgcctg ccaaacagac atgatgctgt tt 1002
<210> 97
<211> 1257
<212> ДНК
<213> Cricetulus griseus
<220>
<221> misc_feature
<223> Rad52
<400> 97
atggctggag ctgaggaaac agtccgtgga ggctgtgaca cccatcctcc ctttgctggt 60
gggaaatctg tgctgtgctt tgggcagagc cagtacacag cagaggaata ccaggctatc 120
cagagggctc tgaggcagcg gctgggcccg gagtacatca gcagccgcat ggctggagga 180
ggccagaagg tgtgttatat tgaaggtcat cgagtaatta acctggccaa tgagatgttt 240
ggttacaatg gctgggcaca ctccatcacc cagcagaatg tggattttgt tgacttcaac 300
aatggcaaat tctacgtggg agtctgtgca tttgtaaggg tgcagttaaa ggatggttcc 360
tatcatgagg acgtgggtta tggagttagt gagggcctaa ggtcaaaggc cttgtcactg 420
gagaaggcca ggaaggaggc tgtgactgac gggctgaagc gggcactcag gagttttgga 480
aatgcacttg gaaactgcat tctggacaaa gactatctga ggtcactaaa taagcttcca 540
cgacagctcc ctcttgaagt ggatttaact aaagcaaaga gagaagattt tgaaccatct 600
gtggaacagg caagatataa tagctgccta cagaatgaag caccgggacc cccaaaacca 660
caagaagcgg cttccccttg cagaccaagc cacccacatg attcgaacat taggctgcag 720
ggggctaagg actgcagcag ctcctgcagt ctggccgccc ccatggagag tgatgccatt 780
caccagcgca agctccggaa gctccggcag aaacagctgc agcagcagtt ccggcagcag 840
atggaggccc acctacaggg ccacacacct gccgtaaaag tgaaagccga gcgtgaggca 900
gtgcttccag accttcctcc aaaacacagt acccctgtaa ctgctgcctc agaactcctc 960
agggagaaag ccatttttcc agataaccct gaagacaacc ttgaaatgtg ggacctgact 1020
ccggatttag aggacatcat taagcccttg tctagaccag aaccacctca aacctctgcc 1080
accagagtcc aggtgatcca ggatggtgtc ctacacggcc tttgccacca gatgccacca 1140
gaaaaacatg aagctggtca cctgcaggcc cacagcactc accagcatgt attaggaaac 1200
tctgactctc ataggaagag ccaggacctg aagaaaagga aactggatcc atcctga 1257
<210> 98
<211> 2235
<212> ДНК
<213> Cricetulus griseus
<220>
<221> misc_feature
<223> Rad54
<400> 98
atgaggagga gcttagctcc cagccagttg gccaggagga aaccagaagg cagatcatct 60
gatgatgaag actggcagcc tgggacagta actcctaaga aacaaaaatc cagtaatgag 120
acccagtgct tcctgtctcc ttttcggaaa cctttgactc agttaatcaa ccgaccgcct 180
tgtctggata gcagtcaaca tgaagcgttt attcgaagca ttttgtcaaa gcctttcaag 240
gtccccattc caaattatca aggtcctctg ggctgtcgag cattgggctt gaaaaaggct 300
ggtattcgtc gtgccctcca tgaccctctg gaagaaggtg ccttggttct gtatgagcct 360
cccccactaa gcgtccatga ccaactgaag ctggacaagg agaaactccc tgtccacgtg 420
gttgttgatc ctattctcag taaggtgttg cggcctcatc agagagaggg agtgaagttc 480
ctatgggagt gtgtcaccag tcgtcgaatc cctggaagcc atggctgtat catggctgat 540
gagatgggcc tgggaaagac actacagtgc atcacattga tgtggacact tttacgccag 600
agcccagagt gcaagccaga aattgagaaa gcagtggtag tgtcaccttc cagcttggtg 660
aagaactggt acaatgaggt tggaaagtgg cttggaggca ggatccaacc tctggccatc 720
gacggaggct ctaaggacga gatagaccga aaactggaag gattcatgaa ccagcgtgga 780
gcgagagtgc cttctcctat tctcatcatt tcctatgaga cttttcgcct tcatgtcgga 840
gtccttaaaa aaggaaatgt tggactggtc atatgtgatg agggacacag gcttaaaaac 900
tctgagaatc agacttacca ggccctggac agcttgaata ccagtcgtcg ggtgctcatc 960
tctgggaccc ccatccaaaa tgacttgctt gaatatttca gcttggtgca cttcgttaat 1020
tcaggcattt tgggaactgc ccaggagttc aagaagcatt ttgagctgcc aattttgagg 1080
agtcgagatg cagctgccag cgaggcagac aggcagctag gggaggaacg tcttcgagag 1140
ctcatcagta tagtgaatag gtgcctgata cggagaacat cagatatcct ctctaaatat 1200
ctgccagtga agattgagca ggtggtttgt tgtaggctga caccccttca aactgagtta 1260
tataagagat ttctgagaca ggctaagcct gaagaagaat tgcgtgaagg caagatgagc 1320
gtgtcttccc tgtcttctat cacctctcta aagaagctat gtaaccatcc agccctaatc 1380
tatgacaagt gtgtgtcagg ggaagatggc tttgaggata ctttggatat cttcccacct 1440
ggttatactt ctaaagctgt agaaccacag ctttcaggta aaatgctggt ccttgattac 1500
attctggcca tgactcgaag ccgcagcagt gataaagttg tgctggtgtc taattatact 1560
caaacgttgg atctctttga aaagctgtgc cgagctcgaa ggtacttgta tgttcgcctg 1620
gatggtacaa tgtccattaa gaagcgagcc aaggttgtgg agcgcttcaa tagtccatcg 1680
agtcccgatt ttgtcttcat gctgagcagc aaagctgggg gctgtggact taatctcatt 1740
ggtgctaacc ggctggtcat gtttgatcct gactggaacc cagccaatga tgaacaagct 1800
atggcccgag tctggcgtga tggtcaaaag aagacctgct atatttaccg actgctatct 1860
gctggaacca tagaagagaa gatctttcag cggcagagcc ataagaaggc actgagcagc 1920
tgtgtggttg atgaggagca agatgtagag agacatttct ctcttggcga gctcaaagag 1980
ctgtttaccc tggatgaagc tagcctcagc gacacacatg acaggctgca ttgccgccgt 2040
tgtgtaaaca gacgccaggt ctggccaccc cctgacggtt ctgactgtac ttcagatctg 2100
gctcagtgga accatagcac agataaacgg ggactccagg atgaggtact ccaggctgcc 2160
tgggatgctt cacctacagc catcaccttc gtcttccatc atcgttctca tgaggagcag 2220
cggggtctcc actga 2235
<210> 99
<211> 837
<212> ДНК
<213> Cricetulus griseus
<220>
<221> misc_feature
<223> Xrcc2
<400> 99
atgtgcagcg actttcgtaa ggccgagtcc gggacggagc tccttgcccg gcttgaaggc 60
agaagctccc tgaaagaact agaacccaac ctgtttgctg atgaagattc accagtacat 120
ggtgatgttc ttgaatttca tggtccagaa ggaacaggaa aaacagaaat gctttatcat 180
ttaacagccc gatgtatact tccaaaatca gagggtggac tgcaaataga agtcttattt 240
attgacacag attaccactt tgacatgctc cggcttgtga cagtgctcga gcacagactg 300
tctcgaagct ccgaggagac catcaagctc tgcctgggaa gattgttcct ggcctactgc 360
agcagcagct tgcagctact gctcacgctg cactcactgg aagccctgtt ctgtagtcac 420
ccctctctct gccttctcat tgtggatagc ctgtcggctt tttactggac agaccgcgcc 480
agtggaggag agagtgtggc cctgcaggaa tccactctga agaagtgttc tcagctccta 540
gagaggcttg tcactgagta ccgcttggtg cttttcacaa caacacaaag tctaatgcag 600
aaagcctctg actcagcgga gcagcctgct tcctccaagc tcccaggtga cggagacaca 660
gactacagag cctatctctg caaggcctgg cagaaggtgg tgaagcacag agtcatcttc 720
tccagagagg acgaggctaa gagcagccgc ttctcattag tttcacgtca tttaaaaagt 780
aacagtttaa aaaaacatgc ttttatgatc agagaaagtg gggtggaatt ctgttga 837
<210> 100
<211> 1050
<212> ДНК
<213> Cricetulus griseus
<220>
<221> misc_feature
<223> Xrcc3
<400> 100
atggacttgg atcaactgga cctaaatccc agaattactg ctgcaattaa gaaggggaga 60
ctgaggtcag tgaaggaggt tctgtgctac tcgggaccag acctgcagag gctcaccagc 120
ctgtccaccc acgatgtgca gcacctactg agagtggccg ctctgcacct ccagggcagc 180
cgggtcctca cagcactgca gctgttccag cagagggaga gcttccccga gcagcatcaa 240
cgcctgagcc tgggctgccc ggtcttggat cagttcctgg gtggcggcct gcccctggag 300
ggcatcactg acctggctgg tcgaagctct gcagggaaga cccagctggg gctacagctc 360
tgcttgactg tgcagttccc acgacaatat ggaggcctgg aggctggggc tgtctacatc 420
tgcacagagg atgccttccc cagcaagcgg ctgtggcagc tcattgaaca gcaacagcag 480
ctgcggacgg atgttcctgg ggaggtgatc cagaagatca gattcagcaa ccacatcttc 540
atcgagcatg cggccgacgt ggacgccttg ctggagtgtg tgagcaagag ggttcccatt 600
ctgctgtcaa gggggatggc ccgcctggta gtggttgact ctgttgctgc cccattccgt 660
tgtgagtatg atgctcaggc cttggccacc agggccaagc acctgcagtc tctgggagcc 720
gcgctccgca gactgagcag taccttccgg agccctgtgc tgtgcatcaa ccaggtgatg 780
gaaacggtgg aggagcaaga gtctatgccc aggccactgg gggcctggga tgagcacctc 840
tctccagccc ttggcatcac ctgggccaac cagatcctga tgagactgat ggttgaccgg 900
gcacatgagg acgatgcctc catgggctta cccagaagcc cggcacggac catacgggtg 960
ctctctgccc cgcacctgcc cctctcctcc tgctgctaca cggtcagtgc ggaaggcatc 1020
agagggatac cgggaaccga gtcctgctaa 1050
<210> 101
<211> 5373
<212> ДНК
<213> Cricetulus griseus
<220>
<221> misc_feature
<223> Brca1
<400> 101
atggatttat ctgctgttca aatcgaagaa gtacaaaatg tccttcatgc tatgcagaaa 60
atcctggagt gtccaatctg tttggaattg atcaaagaac ccatttccac aaagtgtgac 120
cacatatttt gcaaattttg tatgctgaaa cttcttaacc agaagaaagg gccttcacaa 180
tgtcctttgt gtaagaatga gataaccaaa aggagcctgc aaggaagcac gaggtttagt 240
cagcttgttg aagagctgtt gaaaatcact gatgcttttg agcttgacac aggaatacag 300
tatgcaaacg gttacagtat ttcaaaattg aaaaattctt ctgaaccttt gaatgaggaa 360
gcttccatca tccagagtgt gggctaccga aaccgcggca aaagacttag acagattgaa 420
tctggaaatg ccaccttgaa ggacagtctc agtgtccagc tgtctaacct tggaattgtg 480
agatcaacaa agaaaaatca tcagacacag cctcgaaata aatctgtgta cattgaatta 540
gagtctgatt cttctgaaga gacagttagt aaaccagatg actgcagtgt gagagaccag 600
gaactgttac aaaccacctc tctgggagct ggagatgaag tcagtttgga ttcttcaacc 660
agagctgctt gtgagttttc tgaggacgta acaaatattg atctgcacca gtgcagtaat 720
aaagatttgg accccattga gaatcatgca actggaagat atccagaaaa atgtcaggat 780
atttctgttt caaacttaca tgtggagcca tgtggcacag atattcatgc cagctcgtta 840
cagcatgaga acagcagctt attactcact gaagacagaa tgaatgtaga aaaggctgag 900
ttctataata aaagcaaaca gtctggctta gccaggagcc aacagaacag atgggctgaa 960
agtaaagaaa catgtaatga taggcagatt cccagcactg agaaaaaggt agatccaaat 1020
actgattcct tctgtgggag aaaaaaatgg aaaaatcaga aaagtctgtg tcctgagaat 1080
tctagagcta cccaggatgt tccttggatg acactgaata gcagcattca gaaagtcaac 1140
gagtggtttt ccaaaactgg tgaaatgcta acttgtgatg gcacatctga caggaggcat 1200
gagtcaaatg ctggagcagc tgttgtgtta gaagtttcag atgaagtaga tggatgttcc 1260
agttcttcaa aagaaatgga cttagtggcc tctgatcccc ataatgcttt aaagtgtaaa 1320
agtgaaagcg acctctccaa accagtagag aacaatatcc gagataaaat atttgggaaa 1380
acgtatcaga gaaagggaag tctccctcac ttgaaccacg taactgaaat tataggcaca 1440
tttattgcag aaccacagat aacacaagaa caccccttca caaataaatt aaaacgtaaa 1500
aggagaacta catgccttca tcctgaggat tttatcaaga aagcagattt aacagttact 1560
caaaagactt ctaaaaatgt aaatcaagga actgaccaaa tggagccaga tggcctagtg 1620
atgggtatta ccagtaatgg tcaagagaat gaaacacaag gtaataatct tcagaaagag 1680
aaaaatgcta atccagtgaa gtccttggaa aaggggtctg tttccacaac taaagccaaa 1740
actataagca acagtataag tgatttggag ctagaattaa atgtccacaa ttcacaggca 1800
cctaagaaaa gtcggctgag gaggaagtct tcaaccagat gtgttcttgc actggaacca 1860
gtcagtagga atccaagccc acctgcttgt actgaatttc aaattgatag ttgtagcagc 1920
agtgaagaaa cagagaaaaa caattccaac ccaacaccag tcaagaacat tagaaagcct 1980
caactcacgg aggacacaga acctgcagca gattccaaga agaataaaga gccaaatgaa 2040
caaagaagga agagaagggc cagtgatcct ttcccagaag agaaatttat gcctggctta 2100
ttaactaact gtccaagttc tagtaaacct caaggacctg ccaatcctag ccctcagaga 2160
aaggaagtag agaaacttga aacaagccaa atgtctgaca gtaccaaaga cctgagggat 2220
ctgttgctgg atggagaaca gggtttgccc actgagaggt cggaggagag taccagtgtt 2280
tcattggtgc ctgacactga ttatgacact cagaacagtg tgtcattgct ggaagctaat 2340
gctgtcagat atgcaaaaac agtatcaagc cagtgtatga cccagtttgt agcaagtgat 2400
aaccctaacg aactggtcca tggttctaaa gatgctggaa gtggcacaga gtgcttcaag 2460
catccattga gacacgaact tagccacatt caggagacca tagaaatgga agagagtgaa 2520
cttgatactc agtatttaca gaatacattt caagcttcaa agcgtcagtc atttacttta 2580
ttttcaaaac caagagatcc ccaagaggag tgtgtaacag cttgtgcttc ctctgtgtcc 2640
ttaagggaca tgagtccaaa agtgacttct gaaggtgaac aacaagaaga aagtcgggga 2700
cacgaagagg ctgaaatcag tcacatacag gcagggcctg cgacagtggg cgtgcctctg 2760
cctcgtcagg aaggtgatcc aggcgctgat acaatgcatg ctgcagtgtg cagcctttgt 2820
ccatcatctc agtacagaag caatgaaaac tcacatccta aacaatcagc ttctcccatc 2880
atgtcttcta taaaaactgg ccatgggaaa accctgtcag aggaacaatc tgagaaacat 2940
acattgtcaa atgaaaaggc aatgggaaat gagacctttg ttcaaagcac aatgcacaca 3000
attagccaaa accacagaaa aaatggttgt caagaagcca actcaggcag tattaatgaa 3060
gtgccttcca gtggtgaaaa cttccaagga cagctaggcc gaaacagaag gactaagtta 3120
aacactgtgc ttccactagg tcttatgcaa cctggagtct gtaagcatag ttttcctgtc 3180
agtgattata aatatcttga aataaaaaag caggaaggtg agaatgttgg tgcacacttc 3240
tcttcgtgtc tgttttcaga taagctcgaa caacctgtag ggagtggtaa tgtttttcag 3300
atttgttctg agacacctga tgacctgttg gatgatgttg aaatacagga aaatactagc 3360
tttggtgaag ttgacataat ggagaagtct gctgttttta acgggagtgt ccagagacga 3420
gagctcagta ggagccctag ccctttaacc catgcatcgc tggctcggaa tctccagaga 3480
cggtctagga agttagagtc ctcagaagac agcggatctt gtgaggatga agaccttccc 3540
tgcttccaac acttacttgg ccaagtaagc aaaacacctg aacctagcag tgttgtgaca 3600
cagcctctgt cagagaaagc agaggggacc caagtgccat ggaagagtag tgttggtgac 3660
tctgataacg aggtgatctt gatagaggca tctcaggaac atcaccctag tgaggatgca 3720
aaatactctg gcagcatgtt ctcttctcag cccagtgctg tacaaggttc aactgcaaat 3780
acaagctccc aggacccctt gtttaacctt tccaaacaaa agagtcacca gtctgaggat 3840
gaggaagatt ttctaagtga caaggaattg atttcagatg atgaggaaat gggaacttgc 3900
ctagaagaag ataatgatca agaagaggat attataatcc cagattcagc tgaagcggca 3960
tctggatatg agagtgaaac aaacctttct gaagattgct cacagagtga tattttaacc 4020
actcagcaga gggccaccat gaaggataac ctgataaagc tccagcagga gatggcccac 4080
ctggaggctg tgctagaaca gcaaggggac cagccttctg tccactcctc gtctctcaca 4140
gctgaacctt gtgcccctga agacctacca aacgcagaac aaaacttatc aggaacagca 4200
attttaactt caaagaatat taatgaaaat cctgtaagcc ggaatccagc atgcgtttct 4260
gctgacaagt tccaaccaca acctccagac agttccagca gtgaaaataa agagtcaagg 4320
gaaggaaggc cttccctttt taaatctccg ttggcaggca gtaggtgctc tgcacacagc 4380
cgctctggga gtcttcaaaa cagaaactgc ccatctcaag aggagctcct ttggactgtt 4440
gaagcaggga agtcagaagg aacgccatac ctggaatctg gaatcagcct tttctctaat 4500
agagaccctg aatctgagtc ccctaaagag ccagcttatg tttgcaccac accagcttca 4560
acctctgcac ggacaatatc ccagtatcag gtttctgact ctttcgagag tccggctgct 4620
actcatgctc atactgcagt gatagaaact gtgagcaaga aaaagccaga attgacatct 4680
tcaaaaggaa gagccaataa aggaatatcc atggtggtgt caggcttgac ccccaaagaa 4740
gtaatgattg tgcaaaagtt tgctgaaaaa taccgcctca ctttaactga tgcaattact 4800
gaggagacta cccatgtcat tatgaaaaca gatgctgagc ttgtgtgtga acggacactg 4860
aaatattttc tgggaattgc aggaggaaaa tggatagtta gctattcatg ggtgattcag 4920
tctattcaag aaagaaaact tctgagtgtg catgaatttg aagtcagagg agatgttgtg 4980
actggaagaa accaccaagg tccaaagcga tccagagaat cccaggaaaa gctcttcaaa 5040
ggcctaacaa tctgttgttg tgagcccttc accaacatgc ccaaagatga gctggagaag 5100
atgctgcagc tgtgtggggc ttccgtggtg aaggagcttt cgtcactcac ccctgacacc 5160
ggtgttcatc caattgtgat tgtacagcca agtgcctgga cagaagagaa tggctgccct 5220
gagattgacc agctgtgtgg ggcgcacctg gtgatgtggg actgggtgtt ggacagtata 5280
tccgtctacc ggtgtcggga tctggacgcc tacctggtac agaatatcac ccatggccac 5340
gacagcagcg agccacagga ccccaatgat tag 5373
<210> 102
<211> 10098
<212> ДНК
<213> Cricetulus griseus
<220>
<221> misc_feature
<223> Brca2
<400> 102
atgtccaatg aatacaaaag gagaccaact ttttttgaaa tttttaaggc acgatgcagc 60
acagcagact taggacctat aagcctgaat tggtttgaag aactttcttc agaagcccca 120
ccatataatt ttgagcctcc agaagaatat gaatataaga ccaacaatta tgagccacag 180
ctgtttaaaa cgccacagag gaagctctct taccatctgt tggcttcaac tccaataata 240
ttcaatgaac aaggtcaaac tctaccagta gaccagtcgc ctttcaaaga actagggaag 300
actcttggaa atagtagaca taaaaatcac cacagaacca aggccaaaat ggactccatg 360
gttgatgttg ccagtccacc tctgaagtct tgtctcagtg aaagccctct tactctgcga 420
tgcacacagg ttgtaccaca aagagaaaag ccagtgatgt gtggaagttt attttctaca 480
ccaaaaatcg aggagggtca gacacctaaa cctatttctg aaagtctggg agttgaggtg 540
gatcctgata tgtcttggtc cagttcatta gctacaccac caacccttag ttccactgtg 600
ctcatagccc gagatgggga agcacgagga attgtgtttc ctgaagactc ccccgctgtt 660
ttgaaaagct acttttccag ccacaatgaa agtctgaaaa agagtggtat atcagttccc 720
tctgtgactg acagcgagaa caaaaaccag agggaagctt ttagtcatgg attggggaaa 780
acgttagagg attcatctgg caaaacaaac agcttcaaag attgccttag gcagtcaata 840
ccaaatgttc tagaagatgg ggagacagct gcagatactt ctgaagaaga tagtttctca 900
ttatgttttc ctaagcataa aaccagaaaa atgcaaaaaa tgagaatggg caagaccagg 960
aagaaaattt tcagtgaaat aagaactgat gaattaagtg aagaaactag aagtggagct 1020
gatggaaaac attcatttgc acttgaaatt gacccaagag atagtgatcc cttagatcca 1080
gatgtgacaa accagaaatg ctttgacaat gggaatgagg aaatctgcga ggaagttgta 1140
cagtcttcag acactcgatg gtctcagcta accctttctc gtctaactgg tacccagaag 1200
ggaaaaatac ctctacctcc tatttcttct tgtaaccaaa ataattcaga aaaagacttc 1260
atagatacga aggaagaagg tattgactct gttactttag aaaattcttt gcctcatatt 1320
tctagttttc cagaaccaga aaagatgttc tgtgagaaaa ctctggtaga taaggaacat 1380
gagggacagc atcttgaatc acatgagaac tccattgcag ggaagcaagt ggtatctgga 1440
acttgtcaag cagcttgcct attgagagag ccacttgaag agtctctggg taatctcttc 1500
tcagagagtg tgactagctc agcctttaca gaagaaccca gtgcctctgc aagtggattg 1560
ggaatatgtg ctgtgtcctc acagagagag gattctttgt gtcctagttc aggtgacact 1620
ggaaactggc catcaactct cactcacact tctgcaactg tgaacaatac aggtttaata 1680
tccagtctaa aaactaaaag aagaaagttt atttactctg tcggtgacag tgcatctcat 1740
caaggaaaaa tactacaggc agacagaaag tcagagctca ctaacacttc tgctcagttc 1800
gaaacaagtg cttttgaagc gccattcacg tttacaaatg ggaattcagg tttatcagat 1860
tcttctctcc aaagaagctg tttacagaat gatgctgaag agccatcttt gtccttatcc 1920
acctcatttg tgactgcttc caagaaagaa agtagttata gtaatgcatt gatatctcag 1980
gatctccatg acaaagaagc aatagtcagt gaagaaaaac tgcagccaca tacagccctg 2040
gaaactgatt gtctgtcgtg cttgccagaa agacaatgtg aatatgatcc aaagggtccc 2100
aaagtttcag atggaaaaga agaagtctta gtctcagcat gtcatcctgc aggacagcac 2160
acagcagcag cacagcccag cagcattagc tttgaattac aggaagaccc tgtcaatggc 2220
cacaatagta caagtcctaa agaaactcct agcttgaagg tgcttctgtc aaagccagtt 2280
gtgctttcta gaggaaaagt gtcatgtaaa atgccagaga aactgcagtg tgagagttat 2340
aaagataata ctgaattaag caaaagcatt cccttgggag gaaataaaat acacatccta 2400
agtgaaaatt ctaaacctcc tgagcttctg ccacctggaa aatatgtaac agaagcatcc 2460
cccacagtga agtctcagtt caatcagaat acaaatctag cagtcaaaaa aaatgaccaa 2520
gaagaaacgc cttttatttc agaagtaaca gtcaatgtga gttctggaga acttttccca 2580
gacaatgaga ataatttcgc ctttcaagta actcatgaaa ataataagac tgccttagga 2640
agtactgtgg aactgcagga agaagacctc cgccatgcta aagggcctaa tctcaacaac 2700
tctcccacag cagtagatgg agacataggt gatgagcaag cagctcatgc actgattatg 2760
gaagactccg attcctcagc tttagtccat gagtgtgcaa agaagagcag aaatactata 2820
gagcagcatc tgaaaggaac cacagacaaa gatttcaatt cctccttgga tgtgaaatca 2880
gatgggaaca atgactatac agacaaatgg ccaggatttt tggatccagt ttttaagcat 2940
aaatttggag gtagcttcag aacagcttcc aataaagaaa taaaactttc agaacataat 3000
gttaagaaaa gcaaaatgtt cttcaaagat attgaagagg agtatcctac tagtttaacc 3060
tgtattgaca tagttaatgc ctcaccatta gcaaaccagg agatactaag tggaccttat 3120
acatttgatt tgcagtcagt cactaccatg tctgcccatc cacagagtca ggcatctgtt 3180
tcttgtgaag atactcacac atcacttcag gtgttacctt caaagcaaga ttttcattcg 3240
aatcacaatt taacacccag ccaaaaggca gaaattacag aactttctac tatcttggaa 3300
gaatcaggaa gtcagtttga attcacacag ttcaaaaagc caagccatgt agcacagaat 3360
aatatacctg aagtgcctgg aaaacagacg gttgccataa atactacttc tgaggggtgg 3420
aaaaggattg gtcttcatct cacagtggat cctgcctctg tagctcagac agatgacagc 3480
aagaaatttg aaggttctgc tggatttaga caaagctttc cttgcctgtt gaaaagcagt 3540
tgtaacaaaa atacatctag ttttttagca aatgtaaatg aaatggagtt tcgaggattt 3600
cgttctgctc ttggcacaaa gcttagtgtg tctagtgagg ctctgcaaaa agctgtgaaa 3660
ctcttcagtg acattgaaag tggtagtgag gagacttcca caaaagtcga cccaagagct 3720
ttgtcttcag gtgctcgtca tgattctggt gcttctgtgt ttaagataag gaaacaaaac 3780
agtggtaaaa gttttgatga gaaaactagt aagtgccaag taacattaca gaataatacc 3840
gaagtgacta ctggtatttt tgttgacaga aatcctgaaa attatgcaag aaatacaaaa 3900
tgtgaagata acaactctac tggttttcaa agaagtcctt ataaattaaa aaactcggag 3960
gatagtgaat caagtacaag cggcacagtt tctgttcatc aagatgatgg tgacttacca 4020
tgtgctgctg atcactgcag caagtaccct gagtcgtgtt cccaatatgt aagggaggaa 4080
aacacacaaa ttaaggaatg tgtatcagat ttaacatgtt tggaagtcat gaaagctgag 4140
gaaacatgct atattcaacc ttcagataaa gaacaattac cttcaggtaa gatggaacaa 4200
aatagaaaag attttaatat atcctttcag actgcaagtg ggaaaaatgt cagagtctct 4260
gaggagtcat taagtaaaag tatgaatatt ttaaatcaag taacagatga atcgatcatc 4320
tcttcagatt ctttgaattc taaatttcat tgtggcacaa ataacaacaa gatgggtatt 4380
tcacatcaca aggaaactac cagtactaaa aaggtatttg aagaacgttt cccagttggg 4440
actgtcagtc aattaccaac tctccagcag catcctagat gtgaaataga aagtatcaaa 4500
gaacctgctc tgttgggttt tcataccgct agtggaaaaa aagtcaaaat tatgcagaaa 4560
tctttagaca aagtgaaaaa cctttttgat gagacacagt atgttagtca tcaagggtca 4620
aaacccttga aggacagaga gaactgcaaa gaaggacttg cattaggatg tgagacaatt 4680
gaaatacctg cctccaagtg tgaagaaatg cagaagagct ttctctctaa ggagtctgaa 4740
gtgctatcta agcaaagtga tcgtttgtat aggtcgactg aaaatctcag aacatcaaat 4800
ggtacctctt ctaaagctaa tgtacatgga aacatagaaa gtgaaataga aaaaagtcct 4860
acaacttgct gcattagtca tttatcttat tcagtcactg aagattctgc tttgacatgt 4920
gacacaggac acggtagaaa aacttgtgtc agtgagtctt ctctatccaa agacagaaaa 4980
tggcttagag accgactggg tgataagctt caaaaaagag atgctgccga aattgaatgt 5040
gtaaaagaac atactgaggg ttatgctgga gatgcctcat gtgagcatag tttagacagt 5100
atcagaaatg aagttgatat aaattgtgtc tctgaaaatc aaacttcagc cctctttagt 5160
gaccctagca tgtgtcatag ctgtccatcc cattttggtt gtcattgtga taacaagcat 5220
aatgactcag gatatttctc aaaaaataaa atttattctg atactcagcc agacacgaag 5280
aatgaagaca ctgccaattt ttccagtgta tatgctacaa aagaagtaaa tatatacccg 5340
ccaactgtaa atgaagatat ttgtgttcag aaacttgaga ctaactcttc accacataca 5400
aataaaaatg tagccattga cttggctata gcagattcaa ggaattgtaa ggtatgccca 5460
tccaagttca ttacagatca ctcacaagaa actgtgaaaa cagtaaaagc aatatttaca 5520
cataacagtg ataaaacaat taagcaaaac acaaagagta aaccagatac ttgccggaca 5580
agctgtcaga aagcattgga taattcagag gattttatat gtcctagctc tttagaagat 5640
gactatatga actcacataa gacttctgtt tatacccacg atgaacaaat attacagcat 5700
aacctaagtg tgtctggact ggagaaagct caaataccac ctgttcactt ggaaacttgg 5760
gataagtgta aatctacaag ggaacttgca caggcagcct gttcttcaca catgccgggg 5820
atttttagca cagcaagtgg aaaagctgta caggtatcag acgcttcatt agaaaaggca 5880
aggcaagtgt tttctgagat ggatggtggt gctaaacagt tactttccac attgtctctg 5940
gaaagtcatg aacaatcaga ccactctggg agaagagaaa actctgtgac acataaccct 6000
gaggatgtat tgtcactccc aaaaaccttt gcaagcaatg ccaattcatc tgtattctct 6060
ggatttagta cagcaggtgg taaacgggtc acagtttcag aaagtgcctt acacaaagtt 6120
aagggaatgt tagaggaatt tgatttgatc ggaactgaac atactctcca gtgtccacct 6180
acatctgaag gtgtatcaaa aatacttcct caatattgtg ttgaaaagag aaccccagaa 6240
taccctataa actctaaatt gcagaaaacc tatgatgata aattcagttt accaaacaac 6300
tataaagaaa gtgcttcttt ggggaatact cattctcttg aagcttctcc ccaactctct 6360
cagtttaagc aagacacacg gttggtatta ggaaccaaag tatcccttct ggaaaaagaa 6420
caaaccttcc ctcaaaatat aaaaacagaa agtggtgtaa tggaaacttg tcctgatgtc 6480
cctgtgagaa caaatgtagg agactgttct gctttcgaca aaaagccaga gaactgtttt 6540
gaaacagaag cggtggagat tgccaaagct tttatggaag atgatgagct gacggattct 6600
gaaccaagtc acgacaaata ctcattgttt acctgccccc aaaatgaggc tttgttaaat 6660
ttgagaacta gaaagagaag aggaatggct gttgatgcag ttggacaacc cccaatcaaa 6720
agaagcttat taaatgagtt tgacaggata atagaaaata aaggaaaatc cttaaagcct 6780
tcaaaaagtg ctccagatgg tacaataaaa gacagaagat tgttcacgca ccacatttct 6840
ctagagccag ttacctgtgg acccttctgc tctaccagag aaaggcaaga aatgcagagt 6900
ccacatttca ctgcacctgc tcaaggactt ctgtctaaag ggcatccttc tgtgcgttcg 6960
gctttggaaa aatcttcaag caattctaca gtttcagtcc agccgactca taaagtttct 7020
gctacaagga atgaaaggac aggatgctca gtcacaggca aatcccacaa agtcttcgtc 7080
ccacctttca aaatgaaatc acagtttcac agagataaac atttgaataa caagaatatt 7140
aattcagagg aaaagaacca aaagagcaga gatggagaca gtgaagatgt gaacgacggt 7200
gacattcgtc aatttaaaaa aggcagcttc catcaagagg ccgctagaat tgtcacagag 7260
tgtgaagaag agcctttaga tttaatgaca agccttcaga atgccagaga cctacagaat 7320
atacgaatta aaaagaaaga aaggcatcag ctctgtccac agccaggcag tctgtatctt 7380
accaagtcat ccactctgcc tcggatctct ctgcaggccg cagtaggagg ccggcttccc 7440
tctgcgtgtt ctcataagca gctctatatg tatggcgttt ctaaagagtg cataagcatt 7500
aacagcaaaa atgcggaata ttttcagttt gacattcagg attattttgg taaagaggac 7560
ctgtgtgctg gaaagggcat tcagctggct gatggtggat ggctgatccc ctccaatgaa 7620
gggaaggctg gaaaagaaga attctatagg gctctgtgtg acactccagg tgtggatcca 7680
aagcttattt ctagcctctg ggtctctaat cactaccggt ggattgtgtg gaaactggca 7740
gctatggaat ttgcttttcc taaggaattt gctaacagat gcctaacccc agaaagagtg 7800
ctgcttcaac taaaatacag atacgatgtg gaaattgaca acagcagcag atcagcccta 7860
aagaagatcc tggaaaggga tgatactgct gcaaaaacac tcgttctctg tgtttctgac 7920
gtcatgtcac taagcacaac tggatcagaa acttcaggcg gcaaaactag tggtgcagat 7980
cccaagagca tagacacgat cgaactcaca gacggatggt atgctgtcaa ggcccagcta 8040
gaccctccac ttgtggctct tgtaaagagc aggagactga ctgtgggtca gaaaatcatt 8100
acccaagggg cagaactggt gggatctcct gatgcctgcg cacctctgga agcaccagac 8160
tctcttagac tgaagatttc tgccaacagc acacggcctg ctcgctggca cagcaaactg 8220
gggttctttc atgaccccag gcctttccct ctgcccttgt cctcactgtt cggtgatgga 8280
ggaagcgttg gctgtgtgga catcattgtt cagagagtgt accctttaca gtgggtggag 8340
aagacggcat caggattgta catattccgt aatgagagag aagaggagaa ggaagcagtg 8400
agatttgcag cggcccaaca gaagaaactg gaggccttgt tcaccaaaat ccaggcagag 8460
tttaaagacc atgaagaaga ctcagcgcag cggcgcgtgc tgtcccatgc actcacaagg 8520
cagcaggtct atgctctgca agatggtgca gagctctatg cagcggtgca gagtgcattg 8580
gatccagatc accttgaggg ttgtttcagt gaagagcagc tgagagcctt gaacaaacac 8640
agacaaatgt tgaatgataa gaagcaagca cagatccaat cagaattccg gaaggccgtg 8700
gagtccgctg agcaagagga gggcttatca agggatgttt caactgtgtg gaagcttcga 8760
gttacaagct acgagagaaa agaaaagtca gctctgctga gtatctggcg tccatcatca 8820
gacttacact ccctgttaac agaaggaaag agatacagaa tctatcatct ggcagtatca 8880
aaatctagga gtacatttga acgacctagc atccagttaa cagctacgaa aagaactcag 8940
tatcagcagc taccggctgc cagtgaaacc gtattccagg tttaccagcc gagggagccg 9000
cttgacttca gcagactgtt agagacagcc tttcagcctc cttgttctga agtggacctc 9060
gtaggagctg tagtttctgt tgtaagaaca acaggtcttg ctcctttggt ctacctatca 9120
gacgaatgcc ttaatttatt agtggtaaag tttgggatag accttaatga agacataaag 9180
ccacatgtgc taattgctgc aagcaacctc cagtggcacc cagaatccag atcaggagtg 9240
ccaacgttat ttgccgggaa tctttccata gtttctgcca gtccaaagga ggtctacttc 9300
cagaagcgag tcaacagaat gaggcaaact gttgagaata tcgatacatt ttacaaggag 9360
gcagaaaaga agcttttaca tctgcttaat ggaaacagtc ccaagtggtc caccccaaat 9420
aaagactcta ctcagccggc ctacacttgc cctgcttcag agctccttgc tacaggaggt 9480
cagctaatga ggttctcacc taatagtgag caaagttatc caagtccttt atcacattgc 9540
acaccaaaag aaaagtctac acccctggct cggtcagccc agatggcatc aaagtcttgt 9600
aacagggaga gagagactga tgaccgaaaa acctgcagaa aaagaagagc cttggatttt 9660
ctgagtcggc tgcccttacc tcccccggtc agtcccattt gtacctttgt ctctccggct 9720
gcacagaagg cttttcagcc gccacggagt tgtggcacca aatatgcaac acctatgaag 9780
aaaaaagaac ccagttcccc tcagaggagg acgccatttc agaaggacag tggcatttct 9840
cttctggaac aagattcggt agctgatgaa gaacttgcct tcctcaatac ccaagctctt 9900
gtacctggct caccagaaga aaatcaacaa gtacttcctg gtgactccac aagaacctct 9960
gtgccccaca acaaaagaac ccccagccag cacagaggcc agaccagcat gtaggacccc 10020
gatccaggaa ggggagtcta tctcagggat tacagaggtg gcaagggcag tgatggaaaa 10080
ttagctgtcg agtcttag 10098
<210> 103
<211> 888
<212> ДНК
<213> Cricetulus griseus
<220>
<221> misc_feature
<223> Циклин D1
<400> 103
atggaacacc agctcctgtg ctgcgaagtg gagaccatcc gccgcgcgta ccctgacacc 60
aacctcctga acgaccgagt gctgcgagcc atgctcaaga ccgaggagac ctgcgcgccc 120
tccgtgtctt acttcaagtg cgtgcagagg gagattgtgc cgtccatgcg gaaaatcgtg 180
gccacctgga tgctggaggt ctgcgaggag cagaagtgcg aagaggaggt cttccctctg 240
gccatgaact acctggaccg tttcctgtct ctggagcccc taaaaaagag ccgcctgcag 300
ctgctagggg ccacctgcat gttcgtggcc tccaagatga aggagaccat tcctttgacc 360
gccgagaagt tgtgcatcta cactgacaac tctatccggc ccgaggagct gctgcaaatg 420
gaactccttc tggtgaacaa acttaagtgg aacctggccg ccatgactcc ccacgatttc 480
atcgaacact tcctctccaa aatgccagag gcggatgaga acaagcagat catccgcaag 540
catgcgcaga cctttgtggc cctctgtgcc acagacgtga agttcatttc caacccgccc 600
tccatggtgg ctgccgggag cgtggtggct gcaatgcaag gcctgaacct gggcagcccc 660
aacaactacc tgtcctgcta ccgcacaacg cactttcttt ccagagtaat caagtgtgac 720
ccggactgcc tccgcgcctg ccaggaacag attgaggccc ttctggagtc cagcctgcgc 780
caggcccagc agaacatcaa ccccaaggcc accgaggaag agggagaagc agagggagag 840
actgacctgg cctgcacacc caccgatgta cgagatgtgg acatctga 888
<210> 104
<211> 882
<212> ДНК
<213> Cricetulus griseus
<220>
<221> misc_feature
<223> Ercc1
<400> 104
atggaccttg ggaaagacga gggaagcctg ccgcagccca ccaggaagaa gtttgtgatc 60
ccactggaag acgaggcccc tcctgcaggg gccaagccct tattcagatc ctcacggaac 120
cccagcacca cggccccctc ggtcccagcg gcccctcaga cgtacgccga gtatgccatt 180
gcccagcctc caggaggggc tgggcccaca gggcccacag gctctgaacc tgtgaaggga 240
gagaaccccg gccagacggt gaaaacggga gcgaaatcca atagcatcct tgtgagcccc 300
cggcagaggg gcaaccctgt gttgaagttc gtgcgcaacg tgccctggga attcggcgag 360
gtgacccctg actatgtgct gggacagagc acttgcgccc ttttcctcag cctccgctac 420
cacaatctcc atccagacta catccacgaa cggctgcaga gcctggggaa gagctttgcc 480
ctgcgtgtgc tgttggtcca agtggatgtg aaagatcctc agaaggccct gaaggacctg 540
gctaaaatgt gtatcttagc ggactgcacc ctggtcctgg cctggagtgc cgaggaagca 600
ggacggtacc tggagaccta caaggcatat gagcagaagc ccgctgacct cctcatggag 660
aagctggagc agaacttcct gtcccgggcc accgagtgtc tgaccaccgt gaagtcagtc 720
aacaaaaccg acagccagac cctcctggct acatttggat cccttgaaca gctcttgacg 780
gcatcacggg aggacctagc cttgtgcccc ggcctgggcc cccagaaggc ccgcaggctc 840
tttgacgtcc tccatgaacc cttcctcaaa gtgccccgat ga 882
<210> 105
<211> 4473
<212> ДНК
<213> Cricetulus griseus
<220>
<221> misc_feature
<223> MDC1
<400> 105
atggaaaaca cccaggttat tgactgggat gctgaagagg aggaggagac agaaataccc 60
agtgggtcct tggggtatag cttggagcct atagggcagc tgcgtctctt cagtagtact 120
catggaccag aaagagattt cccactctac cttggcaaga atgtaattgg ccgaagcccc 180
gactgctctg tggccctgcc ttttccatcc atctctaaac agcatgcagt aattgaaatc 240
tcagcttgca acaaagcccc tatcctccag gattgtggga gcctcaatgg cactcaactg 300
gtaaagcccc caaaggtcct aacccctgga gtgagccatc gtctgaagga ccaggaatta 360
attctgtttg cagactttcc ctgccagtac cgtcgcttgg atgtccctcc acctttggtc 420
tctcggggac ttctaactat agagaagacc cccagaatac agggagggtc ccaaacctcc 480
agagttttgt tggctgagga ttcggaggag gaagtagatt ttccttctgg aaggtatgtg 540
gcaaatggat caaggaatcc aacgtctcca tcagcaacag tagtaccaga aagtgatgaa 600
gaggggtctt ctccagccca aagtgtccgt gggccgtctt tgccctttga cttgggcagt 660
gacacagatg aagagctatg tccacagcca gcagttgggg agtcctcttc agttgccagg 720
gatggtgctg ctgcagaggc caagcagctg gaagctaatg gagtgacatc tggtatccag 780
ctggtactgg ctcagcctgc tgagcaaaag ctcagagatg caaaagtcaa gagtgaggct 840
ggcaatggag cggctgtggt tgggaggggc tccactgtgg gtaaggacag caacacggaa 900
atgggtgaag aacaccagcc ttccggcttt gtggacagcg atacagacgt ggaagaagac 960
aggatccctg tgaccccccc tgtagttccg gggaagaagc aagtcctgct tggagttggt 1020
aaaaaggacc ctggagcacc tgtcgtgacc catctgcagg acagcccagc tggtcgtggc 1080
acaggtgtgg aagaaggcaa gactccagtg gctggccctc cagagagaaa ccacacggcc 1140
atggtgatca gcagcgacac agatgaggaa gatggagtct cagcagcagt caccttggcc 1200
catctgaaag acagggggat ggctctgtgg agcagggagc caggcatgga agaggtcaag 1260
tcccaaccac aggtcctcgt agaacgaagc cagagtgcct ctgggaaaga cagtgacaca 1320
gacgtggagg aggaaaagag agaagtggtc cctgacagcc ccatggacac agatgaggct 1380
cttactgtcc cacattcaga gagccaagct ccccatagag ccagtgatga tgtagaccag 1440
ggtgtggata tgggctcccc tggtggtcag ctagagggaa accaggcctc ctctgccaca 1500
gtagaagaca gtagtgcaca agcagggaag gaagtcctgc ctcctgagga ggcctgggaa 1560
acagctgtgg aggaaggctc atcttcagca gcggcagatg taagacaaag caagcagcca 1620
ggagtagaag atgctgggac agagtgggct gcaactgttt gtgagcaggc gagcatgctt 1680
gaggtggggg tccaaggcag gtcacctgct acactggtag agccagtggt ggtgcctaca 1740
gctactctgg gggatcccac tcagccacag agagagggag cccagacccc cacaggaaag 1800
gagagaggag tacaaatggg caggaccaag aatgccaaag actgccgtga tgcagagtct 1860
gaagatgtgt gccttccggc tacccagtgc tttgtagaca gggagggcca gagctcagaa 1920
gctgtccaga gtttggaaga tgagcctacg caagtctttc catgcactct tccccaagag 1980
cctgggcctt cccacctcag cctgcagacg ccaggtccag gtgcccagga tgtgccttgg 2040
gaagtcttag ctacacagcc gttttgtctg agagaagctg aggcctctga actgcagctc 2100
atggacaccc accctgcagc tcatgaatcc cacccatctg tgtctagtgc atcagcagga 2160
cagcagcatc tggttcacac agagccgctg ggaattgaag gcggagagat gcaaactgtg 2220
gagaaagcca tgggccaatt gagttgcaag atggcatctg ctggagagga ctcaaggggt 2280
gatccagaac cctcggccca tcgcctgctt tctccagttc ctgaagcttc ttctccacct 2340
cagagtctgc tcacctctca gagccaaaag ccgtctacac cccagtctct gttacttacc 2400
tctccccctt ctgagctaca ccttcctgaa actcctcaca ccaagcctaa tgtcaggcct 2460
cggcggtcct ccaggatgac ctcctcccca cactcctctg ctgcccttaa gccctatact 2520
acctgcccca caaacctgcc tgctgcccct agaccaacat ctcgggctac tcggggcagg 2580
ggcagggcaa ataggtcctc taccaggacc ccggaaccag ttgtccctac agactctgag 2640
cttcaacctc ccacctccac agaacagtct gtcatcccca aacccacatc tccagtcact 2700
cagggcagca taaatggttc ctttgttaaa acgcctgaac cagttgttct cacaggtccc 2760
aaaatccagc ctcccacctc cacagaacag cctgttaccc caaaccccac acctcaggcc 2820
actcggggca ggccacatag gtcttccatc cagaccccag aaccacttac tcccactggt 2880
cctgacctcc agcctcccac ctccacagaa cagcctgtca tacctaaacc cacatctcgg 2940
gctgctcggg gcaggtcacg taagtcttct accaggaccc cagaaccagc tgtccccact 3000
ggtcctgacc tccagcctcc cacctccaca gaacagcctg tcacacctaa acccacatct 3060
cgggccactc ggggcaggtc acgtaagtct gtcagaaccc cagaaccagc tgtccccact 3120
ggtcctgacc tccagcttcc cacctccaca gctctgggca ctttgggaag ggcatgtaag 3180
tcctccattg aggattctga atcagttgga ccagtagcct ctgattttga acctcccatc 3240
tccacagacc ttgttgcccc tgaggtgaca ggtcagagca taacactaaa gtcttcacca 3300
ctaagtgctt ctccagtttc tgccacctct gaactccagc cacctgtccc cacagcccag 3360
cctgttctcc tggagcccat tcctcaagcc aaccaccgaa ggcggcggaa ggctgctggg 3420
aaacggggct cccacacagt tcccattggc caaaagcctt actctgcacc ctctgaacct 3480
ggttcccaat cttcaatcaa tcaaggcttg gcattggaag ccgctgagtc tattactgtt 3540
gctcctgagc ctgctgtttc ccaggctcca gagacctcca ctcagaatcc cgtggtgcaa 3600
aatgaagcag ctgggagatc agggctcatt cccaagcccc agcctgaggt ttctcgatcc 3660
cgcaagaagc cttctactac cacaacctca ccaattcaaa aacgtccccg aagacaaata 3720
ccccagaaga caatagtccc caaggaagaa gatcctgagg aaatgccagt gaaggaagag 3780
cctcaggaga tagcaattcc aacacccggc aaaagaaaga gggaccgtgt agaggatgag 3840
acccagggaa acccaagtcg gagccggcgg gctaaaccta accaagaagc agcagccccc 3900
aaggtactgt tcacaggagt tgtggattct cggggagagc gtgcagtcct ggctctgggt 3960
ggcagtctag ccagctcagt aaatgaggcc tcccacttgg tcactgatcg catccgaaga 4020
acggtcaagt tcttgtgtgc cctggggaag ggcatcccta tcctctccct gaactggctg 4080
tatcagtcca gaaaggctgg tcacttcctg ccacctgatg actacttggt gactgaccct 4140
gaacaagaga agaactttag cttcagcctt cgggactccc tgagccgggc tcggcaacaa 4200
aaactgctgg agggctatga gatttacgtg acccctggag tgcagccacc cccacctcag 4260
atgggcgaga tcatcagctg ctgtgggggc accaacctgc ccagcatgcc ccagtcctat 4320
aagccttacc gagttgtcat aacttgcact gaagacctac ctcgctgcgc tatcccatct 4380
cgactggggc tgcccctcct ctctcctgag ttcctcctga ctggagtgct aaagcaggaa 4440
gtcacaccag aggcctttgt cctttcaaat tag 4473
<210> 106
<211> 2412
<212> ДНК
<213> Cricetulus griseus
<220>
<221> misc_feature
<223> Bard1
<400> 106
atgcaacttt ctgagtctgt ggttggacta ctaggtaggt acacaagccc tgctcgggaa 60
cgaaacccac gaggagcccc gccccttggc gttgagggcg tggccttggg ccgcgctcct 120
ctcagaaatt cgagcgcgct ctcattgcct ctcccctccc tcttacttgc tataaaggag 180
caacccagga tggtctgctg tctgcttcgg ctcatcatca tccagagtgt cgagttcttt 240
gaaatcatca ctaatattct gaaggagcca gtatgcttag gagggtgtga gcacatcttc 300
tgtagtgtgt gtataagtga ttgcgttgga tcaggatgtc ccgtgtgtta cacaccagcc 360
tggatcctag acctcaagat aaacagacaa ttggacaaca tgattcagct ttataggaag 420
cttcaaaatt tgctacatga caataaactt tcagatccaa aagacaacac atctaggaca 480
ggttcatttg atgatgcaga aagcaagaag aattcaataa aaatgtggtt tagtcctcga 540
agtaagaagg ttagatgtgt tgtgaataaa gtatcagtgc caacccagcc tcaaaagaca 600
aaagatgaca aagcccagga agcctccata tacgaatttg tttccacaag tccccctgaa 660
gttgtttcta agagggctaa aacagcttct agaacatctg caaaaaagca gcagaagaaa 720
tctttagctg agatcaacaa gaaggggaat tcaaggccag aaacagaaga tagcaggttt 780
gattctaaag aggaactgaa agaggagagg gttgtctcct gtagccaaat accagttatg 840
gaaagtccac aggtaaatgg tgaaatagac ttgttagcta gtggttctgt tgcagaatct 900
gaatgttctg gaagattgac cgaagtttct ttaccattgg ctgagcatat agtatctcca 960
ggcactgtga gcaagagtga agagactcct gagaagaaag tctgtgtaaa agatcttctt 1020
tcagtagggc gtaatgaaaa tcacaaatac tgtagcaggc ctcctgatcc tacttctaag 1080
aattgtgaga gaagcattcc gagcaccagc agagatgtca ttaaaccaac agtgcttgca 1140
gaaaatatac tgttggttga ctgttcttca ctgccttcag accagcttca agttgatgtc 1200
acactcagga gaaagagtaa catatcagat aactccctta gcctttcacc aggcacaccc 1260
ccaccactgc tgaataattc aactcccaga caaatgatgt caaaaccctc catagtgaag 1320
ctgtcaccca gcattaccgc caggaaaaga aaccacagag gagagactct gcttcatatt 1380
gcttctatta agggcgatat accttctgtt gaatacctct tgcaaaacgg aaacgacccc 1440
aatgttaaag accatgctgg atggacacca ttgcatgaag cctgcagtca tgggcacctg 1500
aaggtagtgg aattgctgct ccagcataac gctttggtga ataccacagg ctatcagaat 1560
gacacaccac tgcacgatgc agtcaagaac ggccatgtgg atatagtcag ggtgttactg 1620
tccaatggcg cctccaggaa tgctgttaac atatttggtg tacggcctgt ggattataca 1680
gacaatgaaa atataagatc attattgctg ctaccagaga agagtgaatc atcctcaact 1740
agccagtgtg cagttgtgac tgctggacag cgaagaaatg ggcctctggt gcttataggc 1800
agtgggcttt cttcccaaca gcagaaaatg ctcagcaaac ttgagacagt tctcaaggct 1860
aaaagatgtg ctgagtttga cagtacagta actcatgtca ttgttcctga tgatgaagct 1920
caaagtactc tgaagtgtat gcttgggatt ctcaatgggt gttggatcct gaagtttgac 1980
tgggtgaaag cttgtttgca cagcaaagta cgtgaacagg aagaaaagta cgaagttcct 2040
ggaggtccgc agaagagcag gctcaacaga gagcagctgt tgccaaagct atttgatgga 2100
tgctacttct ttttgggggg aaacttcaaa catcatccaa agaatgatct ccttaagctc 2160
attactgcag caggaggcaa ggtgctcagt agaaagccca agccagacag tgacgtgact 2220
cagaccatca acactgttgc ataccatgcc aaccctgact ctgaccagcg cttctgtaca 2280
cagtacatcg tctatgaaga tctgtttaat tgtcgcccag aaagggttcg gcagggcaaa 2340
gtctggatgg ctccttccac ttggcttatc agctgtgtga tggcctttga attgcttcct 2400
cttgacagct ga 2412
<210> 107
<211> 2796
<212> ДНК
<213> Cricetulus griseus
<220>
<221> misc_feature
<223> Lig1 (лигаза 1)
<400> 107
atgagaaaaa aagaaccaga gaggaaaggg gagaactctg ctgccaccat gcagaaaagt 60
atcaggtcat ttttccaacc catgaaagag ggtaaagcac agaagccgga gaaggagaca 120
gctaacagca ccaaagagaa ggagccacct ccaaaggtgg cactgaagga gaggaatcga 180
gcagtgcctg agagtgattc tccagtgaag aggcctggaa ggaaggcagc ccaggtccta 240
agcagcgaag gggaggagga agatgaagcc cccagcaccc ctaaagtcca gaagtctgtg 300
tcagactcca aacaaagctc tcctcccagc cctgacgcat gtcctgagaa cagtcctttc 360
cacagtagcc cctccatgga gatctcccca tcaggattcc cgaagcgtcg cactgctcgg 420
aagcagctcc cgaaacggac aattgaggac actgtggagg agcagaatga ggacaaaggc 480
agagcagcca agaaaaggaa gaaggaagaa gaagcacaga ctccaatgga aagcctcaca 540
gagagtgaag atgtaaaacc caaggaagaa aaggaggagg gcaagcatgc tgaggcttcc 600
aagtcccctg agtcgggaac cttgacaaag acagagacca tcccagtgtg taaggccggc 660
gtgaaacaga agcctcagga agaggagcag agcaagcctc ctgccagagg cgccaagaca 720
ctcagcagct tcttcactcc ccggaagcca gcagagaaag ccatagtgaa acaagaagag 780
ccaggtactc cagggaagga agagaccaag ggagccctgg atccaacaaa ttacaatcct 840
tccaagagaa actaccaccc cattgaagat gcctgctgga aacatggcca gaaagtccct 900
tttctcgctg tggcccggac ctttgagaag attgaggagg tttctgctcg gctcaagatg 960
gtggagacac tgagcaactt gctgcgctcg gtggtggccc tgtcacctcc agacctgctt 1020
cctgttcttt acctcagcct caaccgcctt gggccacctc agcagggact agagctgggt 1080
gttggtgatg gtgtcctcct taaggcagtt gcccaggcca caggccgtca gctggagtcc 1140
atccgggctg aggtagctga gaagggtgac gtgggactgg tggccgagaa cagccgcagc 1200
actcagagac tcatgctgcc ccctcctccg ctcaccacct ccggggtctt taccaaattc 1260
tgtgacattg cccggctcac tggcagtgct tccatggcca agaagttgga tgtcatcaag 1320
ggcctgtttg ttgcctgccg tcactcggaa gcccggttca ttgccaggtc cctaagtgga 1380
cgcctgcgcc tcgggctggc tgagcagtcc gtcttggctg cccttgccct ggctgtgagc 1440
ctcacacccc ctggccaaga atttccccca gctgttgtgg atgctgggaa gggcaagacc 1500
acagaggcca gaaagacatg gttggaagaa caaggcatga tcttgaagca gaccttctgt 1560
gaggtacctg acctggaccg aatcatcccg gtgctgctgg aacatggcct ggaacgcctc 1620
ccagagcact gcaggctgag cccaggggtc cctcttaaac caatgctggc tcatcccact 1680
cggggtgtca gcgaggtact gaaacgcttt gaggaggtgg actttacctg cgagtacaaa 1740
tatgacgggc agcgggccca gattcatgtt ctggaaggtg gagaggtgaa gatcttcagt 1800
aggaaccagg aagacaacac aggaaagtac ccggacatca tcagccgcat ccccaagatt 1860
aaactcccct cggtcacctc ctttatcctg gacactgagg ctgtggcctg ggaccgggaa 1920
aagaagcaga tccagccatt ccaagtgctc accacacgca agcgcaagga ggttgacgcc 1980
tcggagatac aggtgcaggt gtgtctgtat gcctttgatc tcatctacct caacggagag 2040
tccctgattc gccagcccct gtctcgacgt cggcagctgc tccgggagaa ctttgtggag 2100
acagagggtg agtttgtctt cgccacctcc ctggacacca aggacatcga gcagatcgct 2160
gagttcttgg agcagtccgt gaaggactcc tgtgagggac tgatggtgaa gaccctggat 2220
gttgatgcca cctatgagat tgccaagagg tctcacaact ggctcaagct aaagaaggac 2280
taccttgacg gtgtgggcga cactctggac cttgtggtga ttggcgccta cctgggccgg 2340
gggaagcgtg ccggccggta tgggggcttc ctcttggctg cctatgatga ggagagtgaa 2400
gagctgcagg ccatatgcaa gctgggaact ggattcagtg atgaagagct ggaggagcat 2460
caccagagcc taaaggccct agtgttgccg accccacgcc cctatgtgag gatcgatggg 2520
gcagttgccc cagaccactg gctggaccca aaggtcgtat gggaggtgaa gtgtgcggat 2580
ctctccctgt cccctatcta ccctgctgcg cggggcctgg tggacaaaga gaaagggatc 2640
tcccttcgtt tccctcggtt cattcgtgtc cgtgaagaca agcagccaga gcaggccacc 2700
accagtgacc aggtggcctg tttgtaccgg aagcagagtc agatacagaa ccagcacaac 2760
tcagacttgg actccgactt tgaggactgc tattaa 2796
<210> 108
<211> 2121
<212> ДНК
<213> Cricetulus griseus
<220>
<221> misc_feature
<223> Mre11
<400> 108
atgagccctg cagatccact tgatgatgaa gacactttta aaatcctggt tgccactgat 60
attcaccttg gatttatgga gaaagatgca gttcgaggaa atgatacatt tgtgacactt 120
aatgaaattt taaaacttgc cctggaaaat gaagtggatt ttattttgtt aggtggtgat 180
cttttccatg aaaacaagcc ctcaaggaaa actcttcata gttgcttgga gttgcttagg 240
aagtactgta tgggtgatcg ccctgtgcag tttgagatca tcagtgacca gtcagtcaac 300
tttggtttta gtaggtttcc atgggtgaac tatcaggatg gcaatctcaa catttccatt 360
ccagtgttta gtatccatgg caaccatgac gatcccacag gggcagatgc cctctgtgcc 420
ctggatattt taagctgtgc tgggtttgtg aatcactttg gacggtcaat gtctgtggag 480
aagattgaca ttagtccagt tctgctgcag aaaggaagca caaaacttgc tctgtacggc 540
ctagggtcca ttcctgatga aaggctctat cggatgtttg tcaataaaaa agtaacaatg 600
ctgagaccaa aggaagatga gaactcatgg tttaacttat ttgtgattca tcagaacagg 660
agtaaacatg gaagtaccaa cttcatccca gaacagtttt tggatgactt cattgacctc 720
gttatctggg gccatgaaca cgagtgtaaa attggcccaa ccaaaaatga gcagcagctc 780
ttctatgtgt ctcagcctgg aagctcagtg gtgacttctc tttcccctgg agaagctgtt 840
aagaaacacg tgggcttgct gcgcgttaaa gggaggaaga tgaacctgca gaagctgcct 900
ctccgcacag tgcggcagtt cttcatggag gatgtggttc tcgctaacca cccaaacctg 960
ttcaaccctg acaatcctaa ggtgacccag gccatccaga gcttctgctt ggagaagatt 1020
gaagaaatgc ttgaaaatgc cgagcgcgaa cggctaggga attctcttca accagagaag 1080
cctcttatcc gactacgggt ggactacagt ggaggctttg aaccttttag tgttcttcgc 1140
tttagccaga aatttgtgga tcgggtcgct aaccccaaag atatcatcca ctttttcagg 1200
catagggagc aaaagggaaa aacaggtgaa gagatcaact ttgggaagct tgtttcaaaa 1260
tctccttcgg aaggaacgac actcagagta gaagacctgg tgaagcagta tttccagact 1320
gcggagaaga atgttcagct ctcactgctg acagaaagag ggatgggtga agccgttcaa 1380
gaatttgtgg acaaggaaga aaaagatgcc atcgaggaat tagtgaagta ccagctggag 1440
aaaacacagc ggtttcttaa ggagcgccat atcgatgctc tggaagacaa gattgacgaa 1500
gaggtccggc gtttcagaga aagcagacag agaaatacca acgaagaaga cgatgaagtg 1560
cgagaggcca tgagcagggc ccgggcgctc agatcgcagt cagagaactc tgcctcagcc 1620
ttcagtgctg acgacctgag tttcgatata gcagaacaga cagcaaatga ctctgatgac 1680
agcctgtcag cagtgcccag cagaggccgg ggccgaggcc gaggtcgaag aggaggcaga 1740
gggcagagca ccgcatcaag aggaggatct cagagaggcc gagacactgg gcaggagacg 1800
gctactcgag gcagatgctc aaaggccact acatcgacct ctagaaatat gtccattcta 1860
gacgctttca gatctactcg acagcagcct tccagaaaca ctgccactaa aaattactca 1920
gagactattg aagtggatga atctgatgaa gatgacattt ttcctaccag ttccaaggct 1980
gatcaaaggt ggccaggcac aacatctagc aaacggatgt cccagagcca gatagccaag 2040
ggggttgact ttgaatcaga tgaggacgat gacgatgacc ctttcatgag cagtagttct 2100
ctaagaagcc gaagataata a 2121
<210> 109
<211> 5973
<212> ДНК
<213> Cricetulus griseus
<220>
<221> misc_feature
<223> 53BP1
<400> 109
atgccagggg agcagatgga ccctactgga agtcagttgg attcagattt ctctcagcaa 60
gatactcctt gcctgataat tgaagattct cagcctgaaa gccaggttct ggaagaagat 120
gcgggctctc acttcagtgt gctgtctcga caccttccca atctgcagat acacaaagag 180
aaccctgtgt tggatattgt gtccaatccg gaacaagcag ctgcagaaga gcaaggagac 240
aataatagct ccttcaatga acatctgaaa gagaacaaag ctgctgcaga tcctgtggat 300
tccactcatt tgggcacatg tgattccatc agtcaggtca tagaacagtt gcctcagcca 360
aacaggacaa gcagtgttct gggagtgaca gtggaagctg cttctcttcc agaagaggag 420
aaggaggaag agctggaaga ggagaatgaa gaggtgggag cagatgctgc tgatgccgct 480
ccgtgctccc ttggtgctga agattctgct tcatcacagt tgggctttgg ggttctggaa 540
ctgtcccaga gccaggatgt tgaagaacat acagtgccat atgatgtcaa ccaagaacat 600
atgcagttgg ttaccaccaa ctcgggttct tcccagctgt ctgacatgga tgcaaataat 660
gttaaatgtg aagaacagtc cactgaagat acctccatgg cagaacaacc taacaaagac 720
atccctgtta cagtcgagca cagtaaaggt atccctgtgg tagatgagca aaatctacca 780
ccggcaaggc cagaggatct gccttccagt cctcaagcct ctgctgcagc tgtggaaaca 840
aaggaagagg tacctgccca agagttgcca gaaggggcgc tggaggttca gatgtcctcg 900
gagcctgagg tctcgtccac tcaggaggac ttgtttgagc agagtagtaa aacagcttct 960
gatggttgtt ctactccttc aagggaggaa ggtgggtgtt ctccggtttc cacacccgcc 1020
accaccctgc aactcctgca gctgtctggt cagaggcccc ttgtccagga cagtctttcc 1080
acgaattcct cagatcttgt tgctccttcc cctgatgctt tccgacctac cccttttatc 1140
gttcctagca gtcccacaga gcaaggaggg agaaaagttg agcccttgga tatgtcagtg 1200
atgcctgaag aaggagggga gactttgcag aagcttcagg atgacgaagc agtggagata 1260
gaaaagcccc atctcccatc tcagccggct gtttccccac aagtgtcaac accagtgtct 1320
cagagcacac ctgtcttcac tccaggctct cttcccatcc cgtcccagcc tgaattttct 1380
catgacattt tcattccatc accaagtttg gaagaaccat caagtgatgg gaagaaaggt 1440
ggcgatttgc acagctcatc tttgactgtg gagtgttcta agacttcaga gagtgaacca 1500
aagaatttca ctgctgatct tgggctctcc ttgacagggg agtcttgcaa actgatgctt 1560
tcttcaagtg agtatagtca atcctcaaag atggagagct tggcttctcc caggagtgag 1620
ggagatggag agaataccca gattgaggac actgaaccgt tgtctccagt caccaattct 1680
aaacttcctg ctgaaagtga tgatgtcctg atgaatccag caccagatga ccaagtagaa 1740
atgaatcaga atgatgacaa agtaaaagag ggtgacacag agaacacagg tgaccatggt 1800
gttttagctg ctggtagtaa aggcagagaa gaaaccgttg ctgaagatgt ttgcattgat 1860
ctcacttgtg attctgggag tcaggcagtt ccgtctcccg ctactcggtc cgaggcactt 1920
tctagtgtct tagatcagga ggaagctatg gaaattaaag aacagcatcc agaggagggg 1980
ttttcgggat ctgaggtaga agaagtcccc gagactccct gtgaaagtca cagagaggag 2040
cccaaggaag aaacgatgga gagtatccca ctgcaccttt ctcttactga aacacagtcc 2100
caggcattgt gtcttcagaa ggaaatgccc aaagaagaat gcccagaggc catggaagtg 2160
gaaccctctg tgattagtgt tgactctccc cagaagctgc cggtactcga ccaggagtca 2220
gagcataagg agccagaggc ctgggaagaa gccgcgtcgg aggactcaag tgtggaggac 2280
tcaagtgtgg ttatcgttga tgtgaaggag ccctcgccaa gagttgtccc ctgcgaacct 2340
ttggagggag cagagaaatg ctcagattcc cactcctggg aggatgtggt gcccgaggtg 2400
gaaccgtgtg ctgcaaatag agtagacact ccggaggaaa agattgtaga atgtgacgga 2460
gatgtgaaag cagagaccac aagaaaggac tctgtagagg aagactcccc acagcctcct 2520
ttgccttcag tgaaagacga gcctcccaga gacgagcctc ccagacccga ccaggagatg 2580
cagcagtccc agcttcaaga gaaagagagc ccagtgacca tagatgcaaa agtggctgat 2640
gccaagcagc tggagtcaga gggagcatcc cagcagcttt cgaaagcccc tgcccgcgac 2700
tcacaaagtt tctgtgaaag ttctagtgaa accccatttc atttcacttt gcctaaagaa 2760
ggtgatatta tcccaccatt gactggtgca accccacctc acattgggca cctaaaattg 2820
gatcgcaaca gacatagtac tccaattggg attggcaact atccagaaag caccatagca 2880
accagtgatg tcatgtctga aagcatggtg gagatcaatg atcctctact tgggagtgaa 2940
aaaggagatt ctgagtctgc cccagaaatg gacggaaaac tgtatctgaa aatgaaactg 3000
gttagtcctg agacagaggc cagtgaagaa tctttgcagt ttagcctgga aaagcctgct 3060
actgctgaga gaaaaaatgg atcaactgct gttgctgagc ctgttgcaaa aaatggatca 3120
actgctgttg ctgagcctgt tgccagtccc cagaagaccg tgcctgtgtt tagctgcagg 3180
caagaggagg tttggagtga ggaccctccc tctgtaccca tcagggcaaa cttgctccat 3240
tttccaagtg ttgaagaaga ggacaaagaa aaactggatg gtaccccaaa gcttaggcag 3300
agtgaacagc ctgtgaggcc cgttgggctg gtcaaggatg ctgctacttc tgaggactct 3360
gcttcttctg ttccccagca gagagcaaca caggggtcat tcagccctca aggagaagtg 3420
atggaaacag acctgctaga aggactgagt gctaaccagg aaaaaccgtg taaggtcttg 3480
atggaaaggc ccacccagag taacatagga atccagacca tggaccattc cctgtgtgct 3540
ccagaaactg tttcagcagc aacccagact gtgaagagtg tatgtgaaca agggaccagt 3600
acagtggacc agaactctgg gaaacaagat gccactgtgc agaccgagag ggggggtgtc 3660
gagaaacagg cccctgtgga cgatacagaa tccctccaca gccagggaga agaagaattt 3720
gaaatgcccc agcctccaca tggccatgtc ttgcatcgtc acatgagaac catccgtgaa 3780
gtccggacac ttgtcacccg tgtcatcaca gatgtttact atgtggatgg gacagaagtg 3840
gaaaggaaag taactgagga gactgaagaa ccaattgtag aatgtcagga atgtgaaaca 3900
gaggtttccc cttcccagac tggaggctct tctggagacc tgggagacat cagttccttc 3960
tcctccaaag catccagctc acaccataca tcaagtggga caagtctctc agccatgcat 4020
agcagtggca gctcaggacg aggagccggg ccactcaaag ggaaaaccag cgggacagaa 4080
cctgcagatt ttgctttacc cagttcccga ggaggcccag gaaaactgag tcctagaaaa 4140
gggatcaatc agacaggggc accagtgtgt gaggaagatg gtgatgcagg ccttggcatc 4200
agacagggag ggaaggctcc tgtcacacct cgtggtcgtg gtcgaagggg ccgcccacct 4260
tctcggacca ctggaacaag agatgcagtt gtgtctggtc cgttgggcat agaagacatt 4320
tcacctagca tgtcaccaga tgacaagtcc ttcacccgaa ttgtgccccg tgtaccagac 4380
tctaccaaac gaattgatac cagttctact gttttgaggc ggagtgattc cccagagatt 4440
ccttttcagg ctgctactgg gtcttctgat ggcttggatg cctcatctcc aggaaatagc 4500
tttgtcgggc tccgtgttgt agccaagtgg tcatccaatg gctattttta ctctgggaaa 4560
atcacacggg atgttggagc tgggaagtat aagctgctct tcgatgatgg gtatgaatgt 4620
gatgtgctgg gcaaagacat tctcctgtgt gaccctatac ccctggacac tgaagtgaca 4680
gccctctcag aagatgagta tttcagtgca ggagtggtca aaggacacag aaaggagtct 4740
ggggagctgt actacagcat tgaaaaagaa ggccaaagga agtggtataa gcggatggca 4800
gtcatcctgt ccttggaaca aggaaacagg ttaagagagc aatatgggct tggcccatat 4860
gaagctgtta cacccctcac aaaggcagca gacatcagcc tagataattt ggtggaagga 4920
aagcggaaac gtcgcagtaa catcagctcc ccagccaccc ccactgcctc cagcagcagc 4980
agcagcagca gcacaacacc cacccgtaaa accacagaga gtccccgtgc ttccacggga 5040
gttccatcag gcaaaaggaa actcatcact tctgaagagg aacggtcccc agctaagcga 5100
ggccgcaagt ctgtcaccgt gaaacctggt acagtggggg caggagaatt tgtgagcccc 5160
tgcgagagtg gagacaacac aggtgaacct tctgtcctgg aagagccaag agggcctttg 5220
cccctcaaca agaccttgtt tctgggctat gcctttctcc ttaccatggc cacaaccaat 5280
gacaagctgg ccagccgctc taaactgcta gatggtccta caggaagcag tgaagaagag 5340
gaggaatttt tagaaattcc tcctttcaac aagcagtata cagaatgcca gcttcgagca 5400
ggagctgggt atatccttga agacttcaat gaagcccagt gtaacacagc ctaccagtgt 5460
ctcctaattg ctgatcagca ttgtcgaacc cggaagtact tcctgtgcct tgccagtgga 5520
attccttgtg tgtctcatgt ctgggtccat gacagctgcc atgccaacca gctccaaaac 5580
taccgtaatt atctgctgcc tgctgggtat agccttgagg agcagcgaat tctggattgg 5640
caacctcgtg aaaacccttt ccagaatctg aaggtactct tggtgtcaga tcaacaacag 5700
aacttcctgg agctctggtc tgagatcctc atgactggag gggcagcctc tgtgaagcag 5760
caccattcaa gtgcccataa caaagacatt gctttagggg tatttgatgt ggtggtgaca 5820
gacccctcat gcccagcctc ggtgctcaag tgtgctgaag ccttgcagct gcctgtggtg 5880
tcacaagagt gggtgatcca gtgcctcatt gttggggaga gaattggatt caagcagcat 5940
ccaaaatata aacatgatta tgtttctcac taa 5973
<210> 110
<211> 384
<212> ДНК
<213> Цитомегаловирус человека
<220>
<221> misc_feature
<223> Энхансер немедленно-ранних генов CMV
<400> 110
gttgacattg attattgact agttattaat agtaatcaat tacggggtca ttagttcata 60
gcccatatat ggagttccgc gttacataac ttacggtaaa tggcccgcct ggctgaccgc 120
ccaacgaccc ccgcccattg acgtcaataa tgacgtatgt tcccatagta acgccaatag 180
ggactttcca ttgacgtcaa tgggtggagt atttacggta aactgcccac ttggcagtac 240
atcaagtgta tcatatgcca agtacgcccc ctattgacgt caatgacggt aaatggcccg 300
cctggcatta tgcccagtac atgaccttat gggactttcc tacttggcag tacatctacg 360
tattagtcat cgctattacc atgg 384
<210> 111
<211> 799
<212> ДНК
<213> Homo sapiens
<220>
<221> misc_feature
<223> Промотор GAPDH человека
<400> 111
gagaagttcc ccaactttcc cgcctctcag cctttgaaag aaagaaaggg gagggggcag 60
gccgcgtgca gccgcgagcg gtgctgggct ccggctccaa ttccccatct cagtcgttcc 120
caaagtcctc ctgtttcatc caagcgtgta agggtccccg tccttgactc cctagtgtcc 180
tgctgcccac agtccagtcc tgggaaccag caccgatcac ctcccatcgg gccaatctca 240
gtcccttccc ccctacgtcg gggcccacac gctcggtgcg tgcccagttg aaccaggcgg 300
ctgcggaaaa aaaaaagcgg ggagaaagta gggcccggct actagcggtt ttacgggcgc 360
acgtagctca ggcctcaaga ccttgggctg ggactggctg agcctggcgg gaggcggggt 420
ccgagtcacc gcctgccgcc gcgcccccgg tttctataaa ttgagcccgc agcctcccgc 480
ttcgctctct gctcctcctg ttcgacagtc agccgcatct tcttttgcgt cgccaggtga 540
agacgggcgg agagaaaccc gggaggctag ggacggcctg aaggcggcag gggcgggcgc 600
aggccggatg tgttcgcgcc gctgcggggt gggcccgggc ggcctccgca ttgcaggggc 660
gggcggagga cgtgatgcgg cgcgggctgg gcatggaggc ctggtggggg aggggagggg 720
aggcgtgtgt gtcggccggg gccactaggc gctcactgtt ctctccctcc gcgcagccga 780
gccacatcgc tcagacacc 799
<210> 112
<211> 1188
<212> ДНК
<213> Homo sapiens
<220>
<221> misc_feature
<223> Промотор Ef1-альфа человека
<400> 112
catgggtgag gctccggtgc ccgtcagtgg gcagagcgca catcgcccac agtccccgag 60
aagttggggg gaggggtcgg caattgaacc ggtgcctaga gaaggtggcg cggggtaaac 120
tgggaaagtg atgtcgtgta ctggctccgc ctttttcccg agggtggggg agaaccgtat 180
ataagtgcag tagtcgccgt gaacgttctt tttcgcaacg ggtttgccgc cagaacacag 240
gtaagtgccg tgtgtggttc ccgcgggcct ggcctcttta cgggttatgg cccttgcgtg 300
ccttgaatta cttccacctg gctgcagtac gtgattcttg atcccgagct tcgggttgga 360
agtgggtggg agagttcgag gccttgcgct taaggagccc cttcgcctcg tgcttgagtt 420
gaggcctggc ctgggcgctg gggccgccgc gtgcgaatct ggtggcacct tcgcgcctgt 480
ctcgctgctt tcgataagtc tctagccatt taaaattttt gatgacctgc tgcgacgctt 540
tttttctggc aagatagtct tgtaaatgcg ggccaagatc tgcacactgg tatttcggtt 600
tttggggccg cgggcggcga cggggcccgt gcgtcccagc gcacatgttc ggcgaggcgg 660
ggcctgcgag cgcggccacc gagaatcgga cgggggtagt ctcaagctgg ccggcctgct 720
ctggtgcctg gcctcgcgcc gccgtgtatc gccccgccct gggcggcaag gctggcccgg 780
tcggcaccag ttgcgtgagc ggaaagatgg ccgcttcccg gccctgctgc agggagctca 840
aaatggagga cgcggcgctc gggagagcgg gcgggtgagt cacccacaca aaggaaaagg 900
gcctttccgt cctcagccgt cgcttcatgt gactccacgg agtaccgggc gccgtccagg 960
cacctcgatt agttctcgag cttttggagt acgtcgtctt taggttgggg ggaggggttt 1020
tatgcgatgg agtttcccca cactgagtgg gtggagactg aagttaggcc agcttggcac 1080
ttgatgtaat tctccttgga atttgccctt tttgagtttg gatcttggtt cattctcaag 1140
cctcagacag tggttcaaag tttttttctt ccatttcagg tgtcgtga 1188
<210> 113
<211> 985
<212> ДНК
<213> Цитомегаловирус обезьяны
<220>
<221> misc_feature
<223> Промотор CMV
<400> 113
gaccatagcc aattcaatat ggcgtatatg gactcatgcc aattcaatat ggtggatctg 60
gacctgtgcc aattcaatat ggcgtatatg gactcgtgcc aattcaatat ggtggatctg 120
gaccccagcc aattcaatat ggcggacttg gcaccatgcc aattcaatat ggcggacttg 180
gcactgtgcc aactggggag gggtctactt ggcacggtgc caagtttgag gaggggtctt 240
ggccctgtgc caagtccgcc atattgaatt ggcatggtgc caataatggc ggccatattg 300
gctatatgcc aggatcaata tataggcaat atccaatatg gccctatgcc aatatggcta 360
ttggccaggt tcaatactat gtattggccc tatgccatat agtattccat atatgggttt 420
tcctattgac gtagatagcc cctcccaatg ggcggtccca tataccatat atggggcttc 480
ctaataccgc ccatagccac tcccccattg acgtcaatgg tctctatata tggtctttcc 540
tattgacgtc atatgggcgg tcctattgac gtatatggcg cctcccccat tgacgtcaat 600
tacggtaaat ggcccgcctg gctcaatgcc cattgacgtc aataggacca cccaccattg 660
acgtcaatgg gatggctcat tgcccattca tatccgttct cacgccccct attgacgtca 720
atgacggtaa atggcccact tggcagtaca tcaatatcta ttaatagtaa cttggcaagt 780
acattactat tggaaggacg ccagggtaca ttggcagtac tcccattgac gtcaatggcg 840
gtaaatggcc cgcgatggct gccaagtaca tccccattga cgtcaatggg gaggggcaat 900
gacgcaaatg ggcgttccat tgacgtaaat gggcggtagg cgtgcctaat gggaggtcta 960
tataagcaat gctcgtttag ggaac 985
<210> 114
<211> 197
<212> ДНК
<213> Вирус 40 обезьяны
<220>
<221> misc_feature
<223> Промотор SV40
<400> 114
tgcatctcaa ttagtcagca accatagtcc cgcccctaac tccgcccatc ccgcccctaa 60
ctccgcccag ttccgcccat tctccgcccc atcgctgact aatttttttt atttatgcag 120
aggccgaggc cgcctcggcc tctgagctat tccagaagta gtgaggaggc ttttttggag 180
gcctaggctt ttgcaaa 197
<210> 115
<211> 999
<212> ДНК
<213> Cricetulus griseus
<220>
<221> misc_feature
<223> Промотор Actb CHO
<400> 115
gggcggaggc cagtcatgca tgttcgggcc tatggggcca gcacccaacg ccaaaactct 60
ccatcctctt cctcaatctc gctttctctc tctctctctt tttttttttt tttttttttt 120
tttttttttt tttgcaaaag gaggggagag ggggtaaaaa aatgctgcac tgtgcggcta 180
ggccggtgag tgagcggcgc ggagccaatc agcgctcgcc gttccgaaag ttgcctttta 240
tggctcgagt ggccgctgtg gcgtcctata aaacccggcg gcgcaacgcg cagccactgt 300
cgagtccgcg tccacccgcg agcacaggcc tttcgcagct ctttcttcgc cgctccacac 360
ccgccaccag gtaagcaggg acaacaggcc cagccggcca cagccctccc gtgggcagtg 420
accgcgctgc agggtcgcgg gggacactcg gcgcggacac cggggaaggc tggagggtgg 480
tgccgggccg cggagcggac actttcagat ccaactttca gtccagggtg tagacccttt 540
acagccgcat tgccacggtg tagacaccgg tggacccgct ctggctcaga gcacgcggct 600
tgggggaacc cattagggtc gcagtgtggg cgctatgaga gccgatgcag ctttcgggtg 660
ttgaaccgta tctgcccacc ttggggggag gacacaaggt cgggagccaa acgccacgat 720
catgccttgg tggcccatgg gtctttgtct aaaccggttt gcccatttgg cttgccgggc 780
gggcgggcgc ggcgggcccg gctcggccgg gtgggggctg ggttgccact gcgcttgcgc 840
gctctatggc tgggtattgg ggcgcgtgca cgctggggag ggagcccttc ctcttccccc 900
tctcccaagt taaacttgcg cgtgcgtatt gagacttgga gcgcggccac cggggttggg 960
cgagggcggg gccgttgtcc ggaaggggcg gggtcgcag 999
<210> 116
<211> 683
<212> ДНК
<213> Cricetulus griseus
<220>
<221> misc_feature
<223> Промотор Hspa5 CHO
<400> 116
acccgagggg acttaggagg agaaaaggcc gcatactgct tcggggtaag ggacagaccg 60
gggaaggacc caagtcccac cgcccagagg gaactgacac gcagaccccg cagcagtccc 120
cgggggccgg gtgacgggag gacctggacg gttaccggcg gaaacggtct cgggttgaga 180
ggtcacctga gggacaggca gctgctgaac caataggacc ggcgcacagg gcggatgctg 240
cctctcattg gcggccgttg agagtaacca gtagccaatg agtcagcccg gggggcgtag 300
cggtgacgta agttgcggag gaggccgctt cgaatcggca gcggccagct tggtggcatg 360
gaccaatcag cgtcctccaa cgagaagcgc cttcaccaat cggaggcctc cacgacgggg 420
ctggggggag ggtatataag ccaagtcggc ggcggcgcgc tccacactgg ccaagacaac 480
agtgaccgga ggacctgcct ttgcggctcc gagaggtaag cgccgcggcc tgctcttgcc 540
agacctcctt tgagcctgtc tcgtggctcc tcctgacccg gggggcttct gtcgccctca 600
gatcggaacg ccgccgcgct ccgggactac agcctgttgc tggacttcga gactgcagac 660
ggaccgaccg ctgagcactg gcc 683
<---
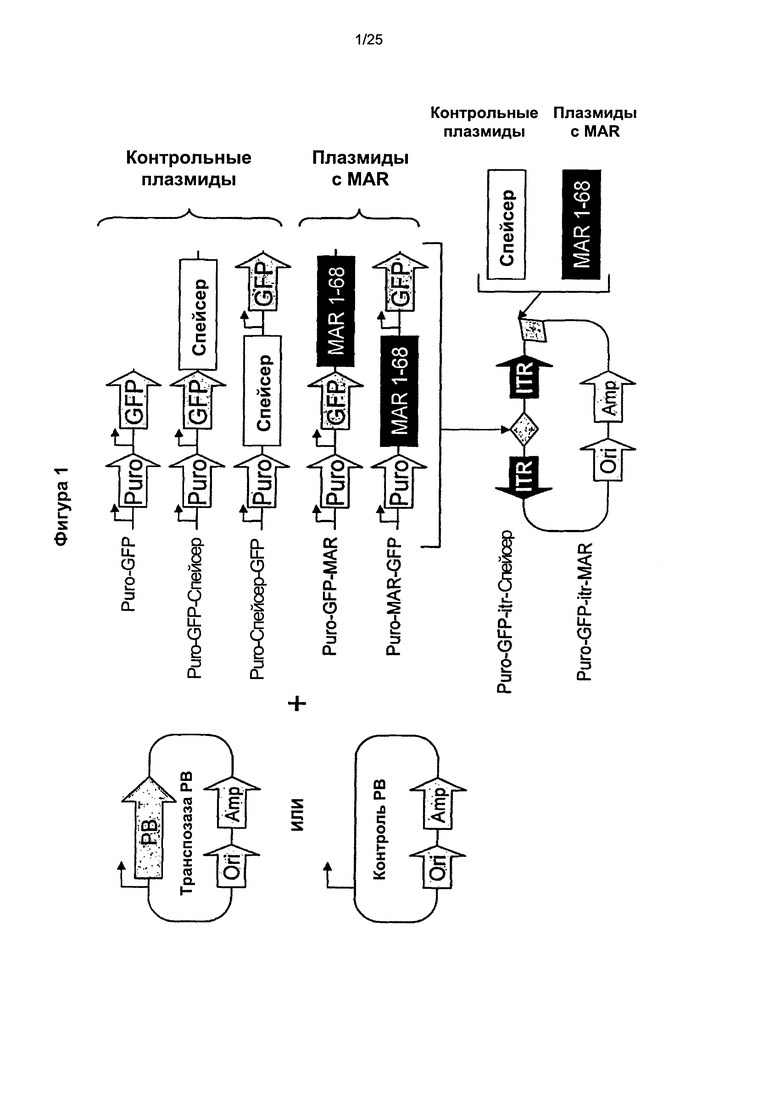
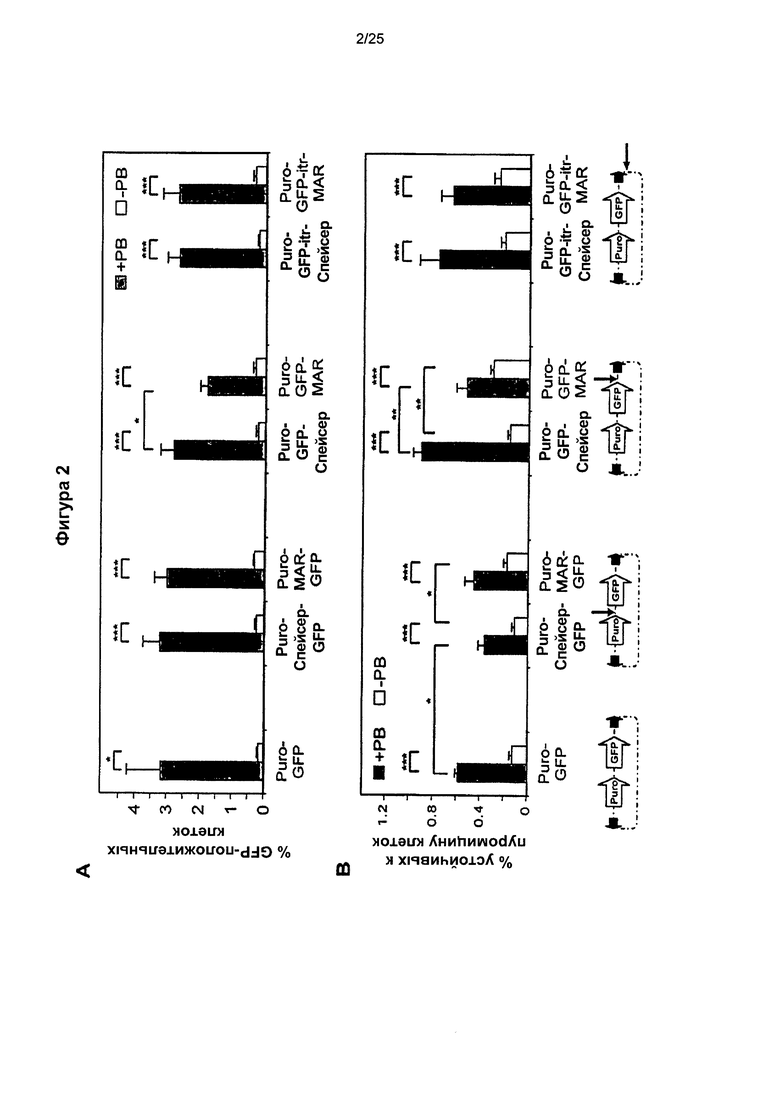
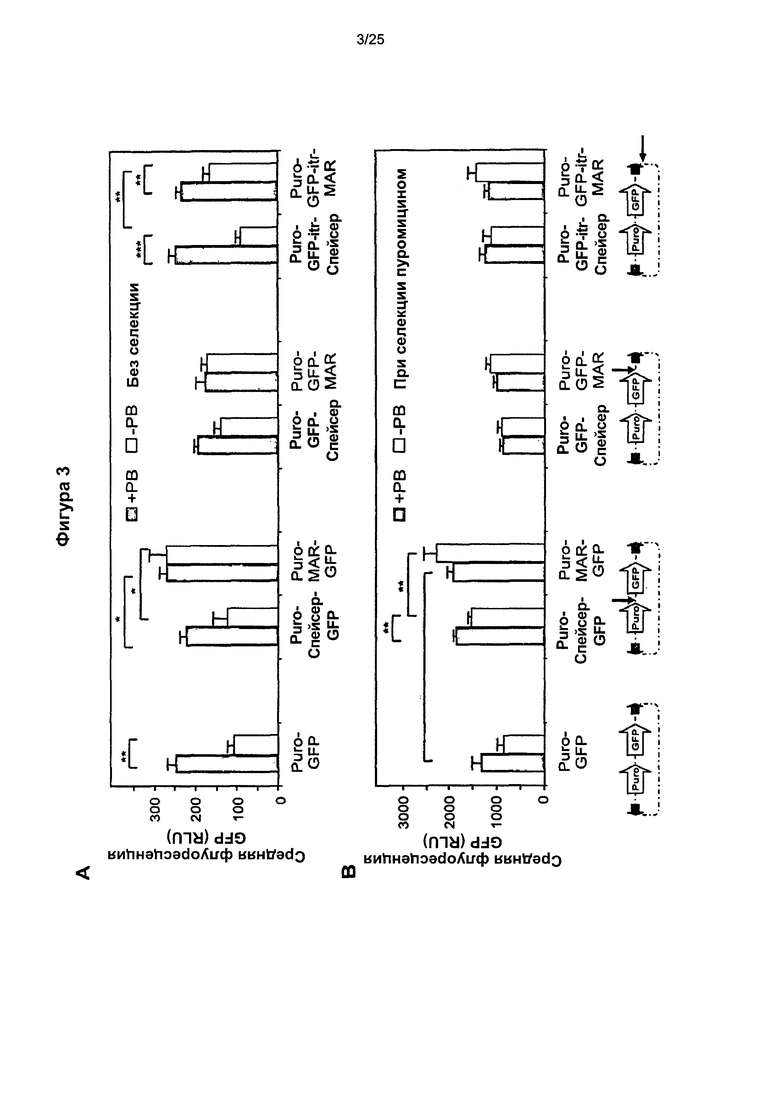
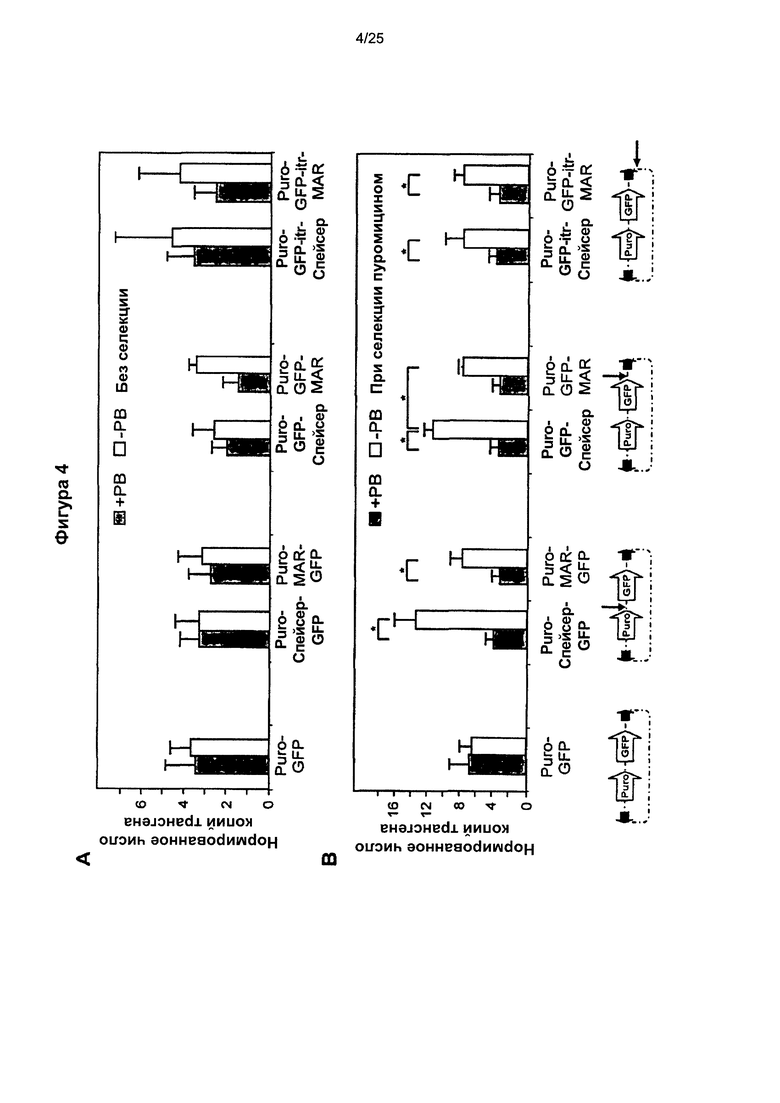
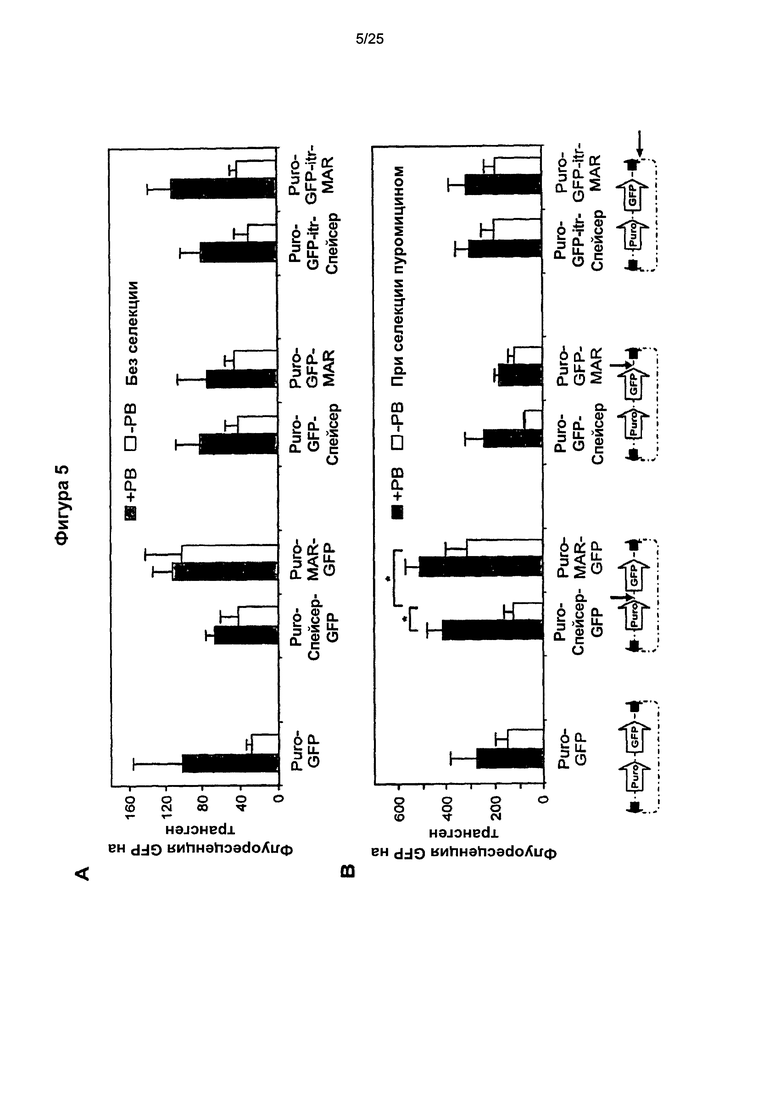
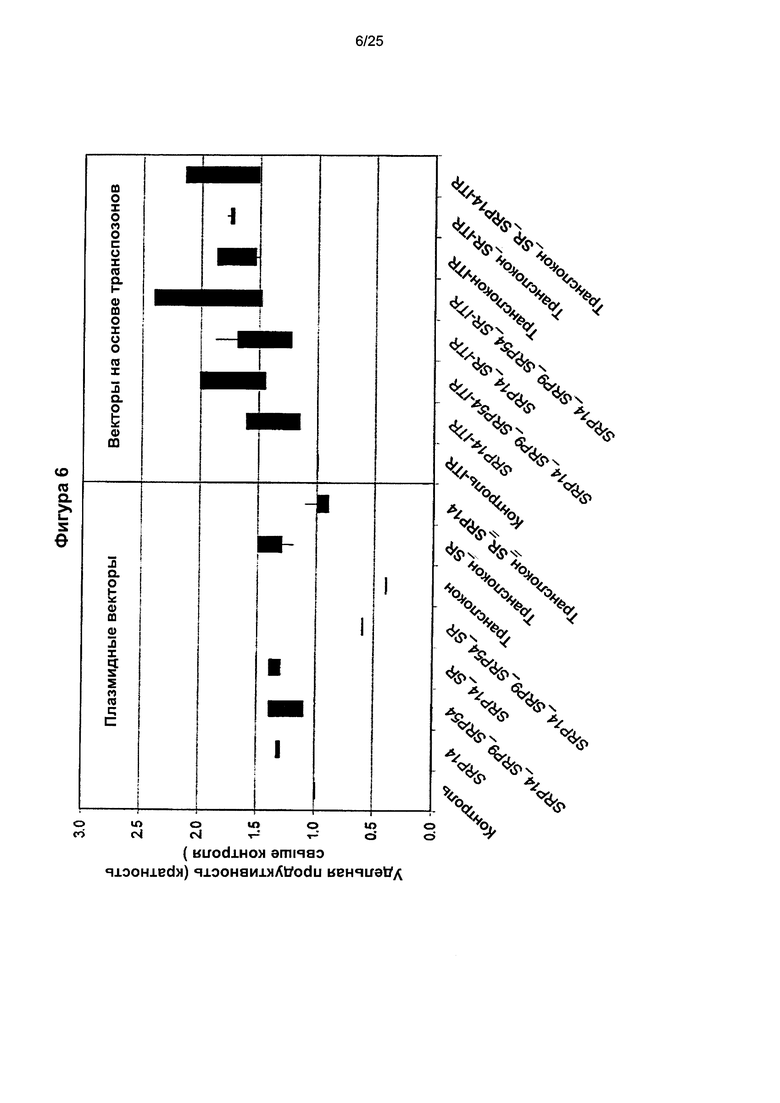
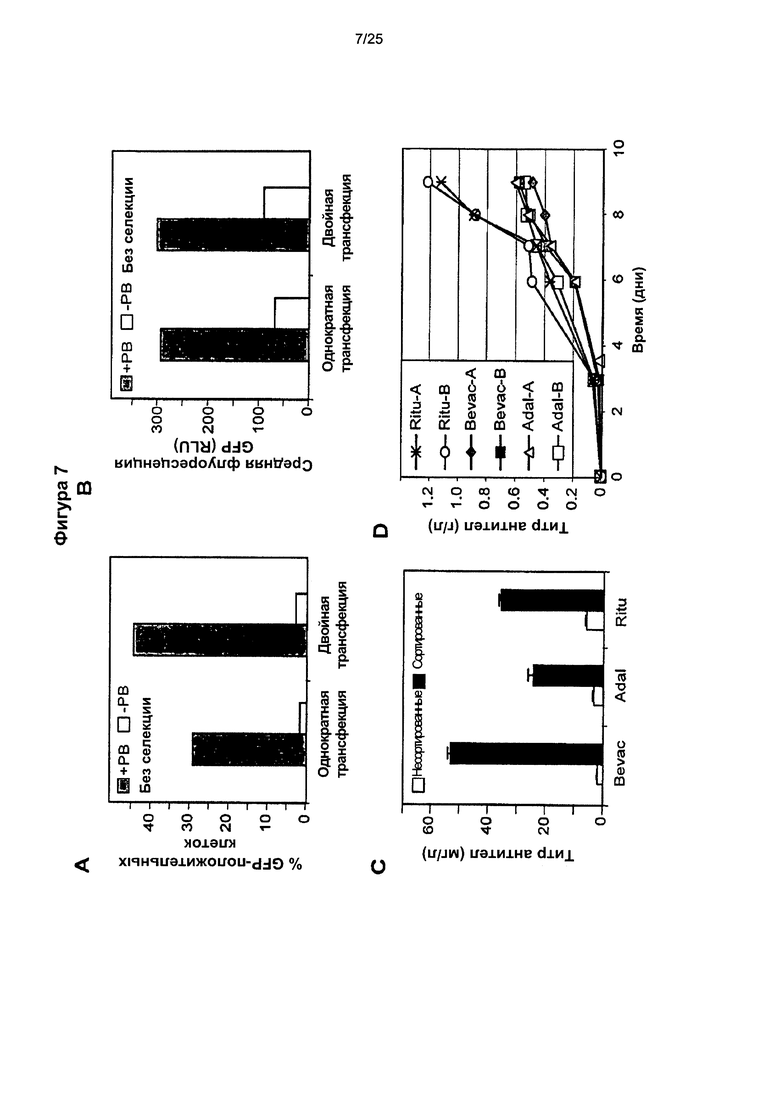
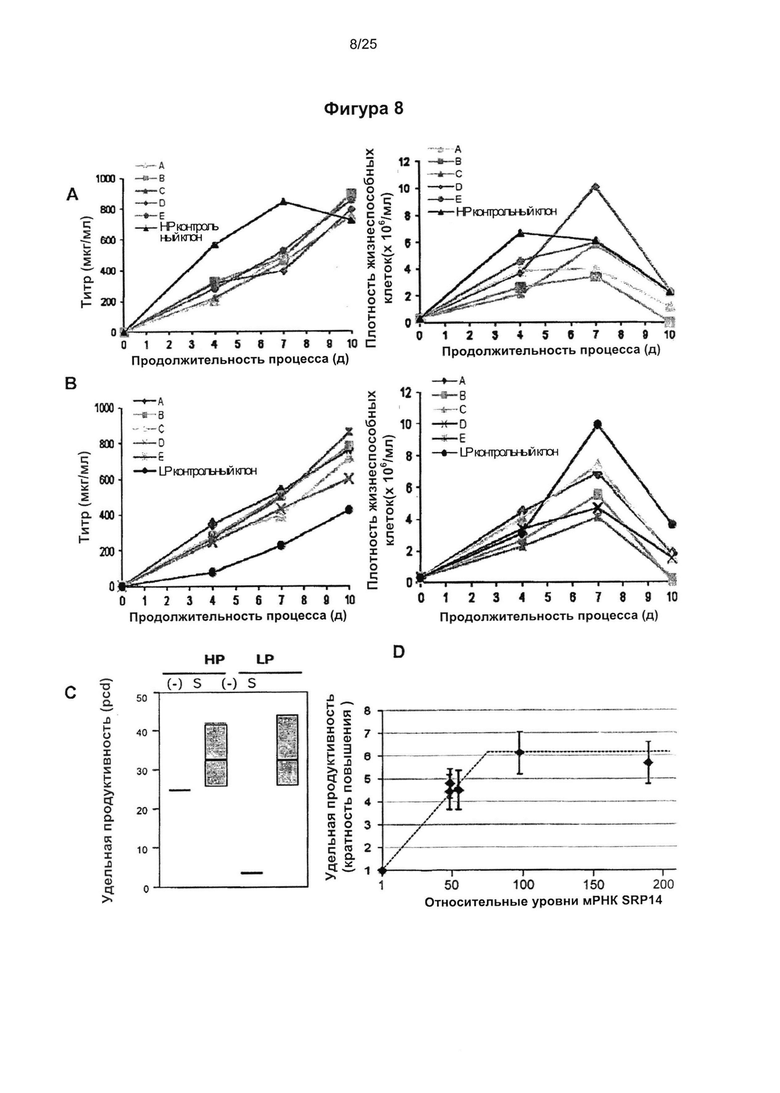
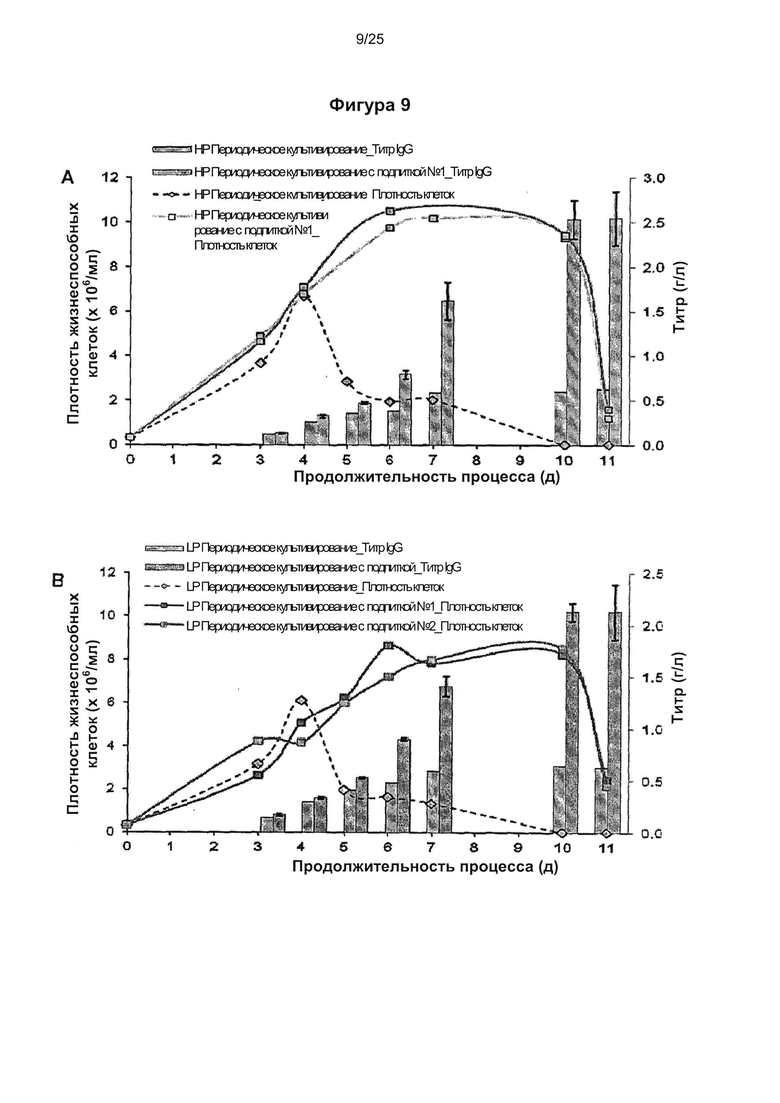

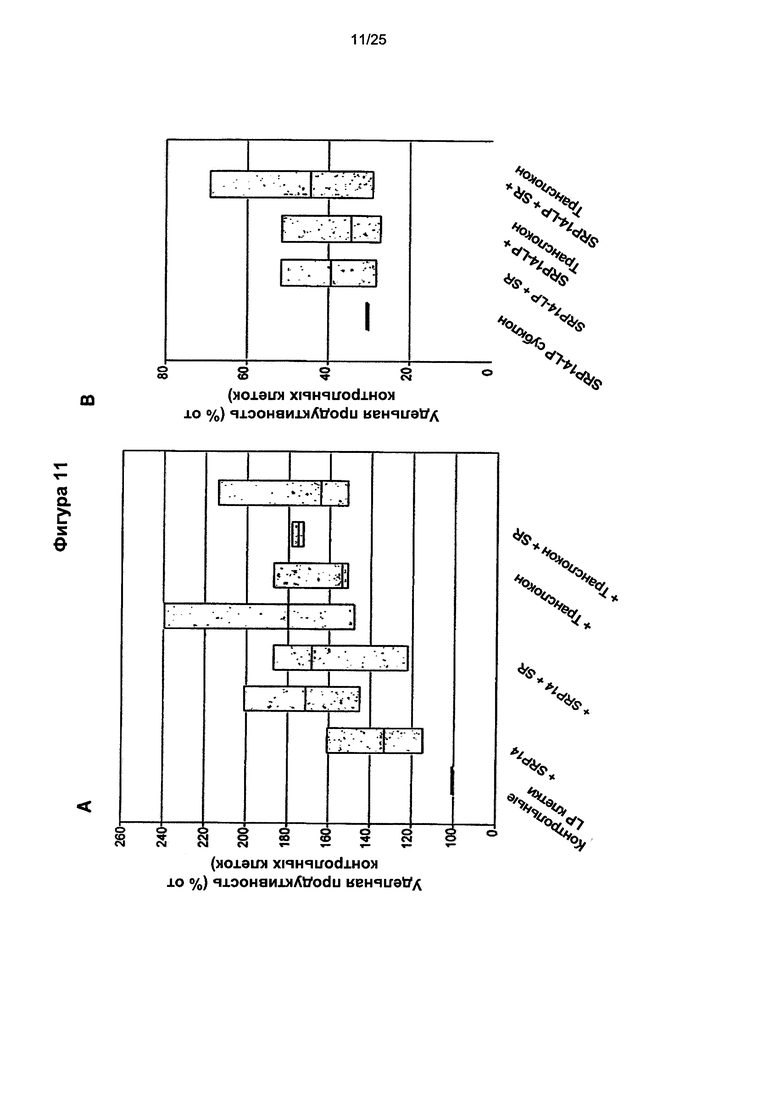
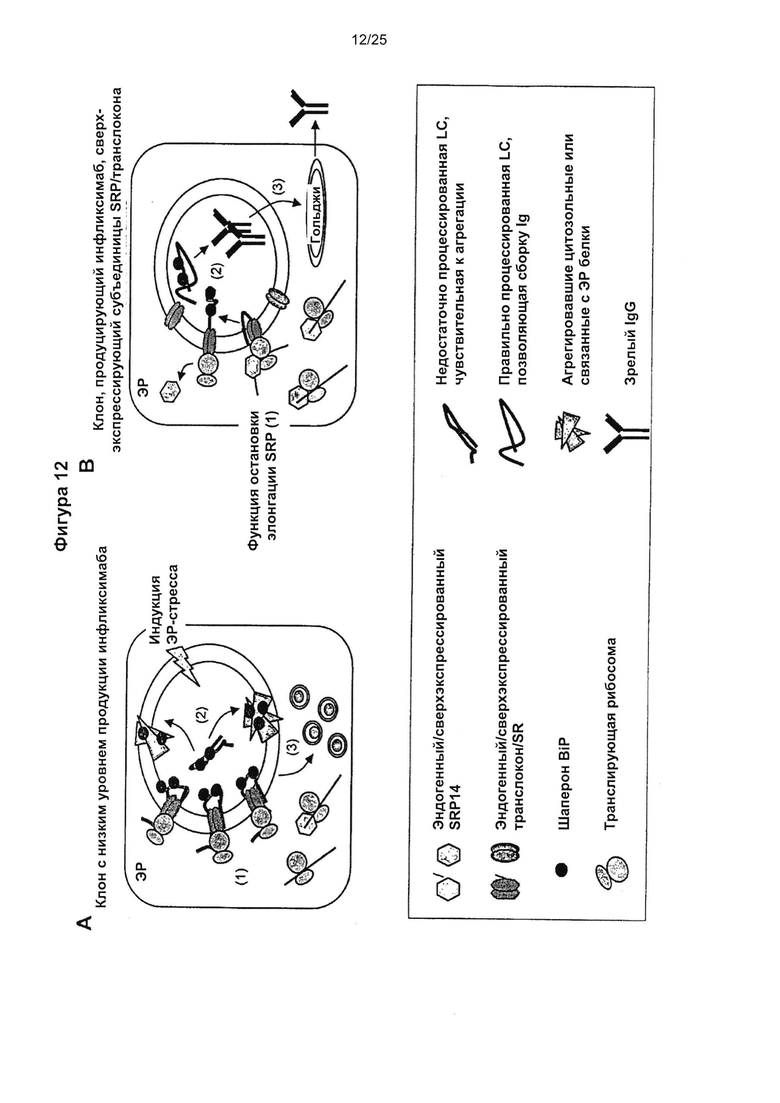
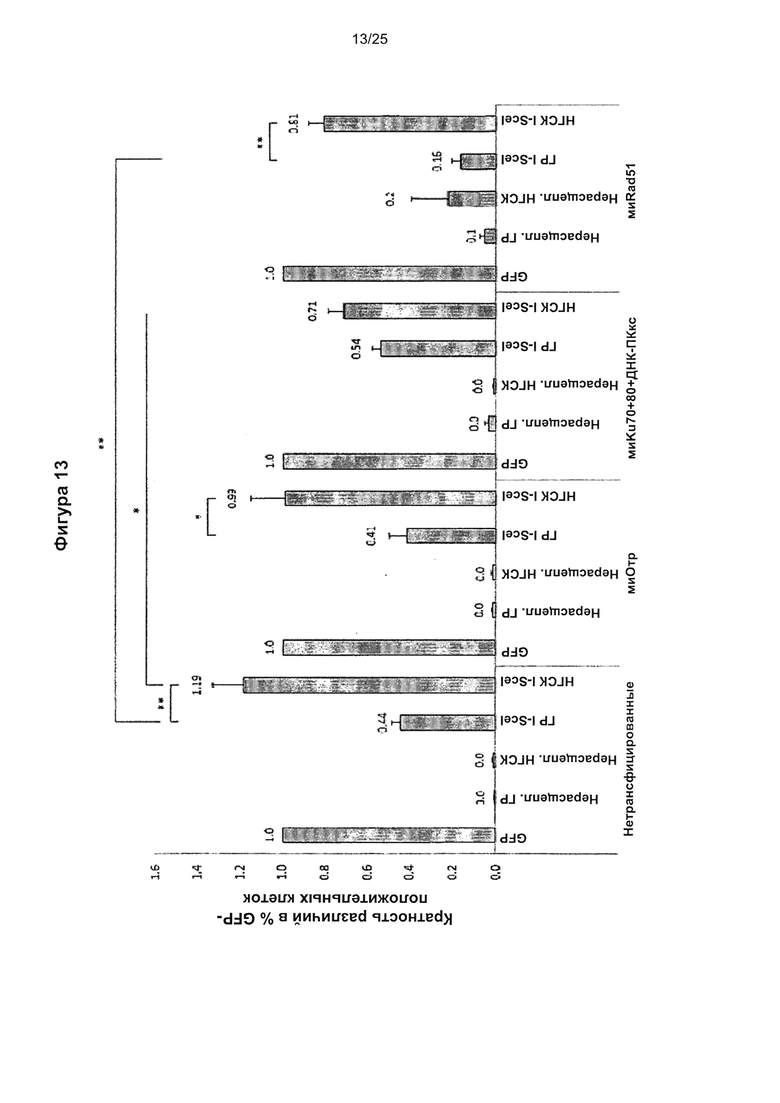
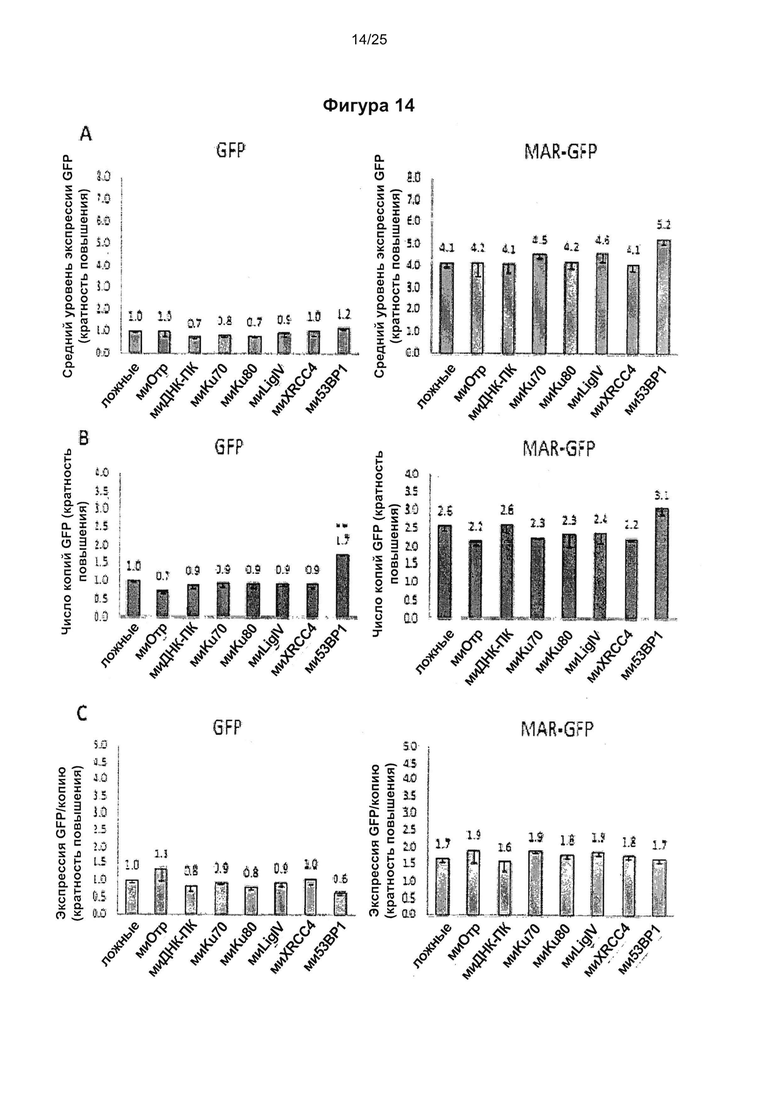
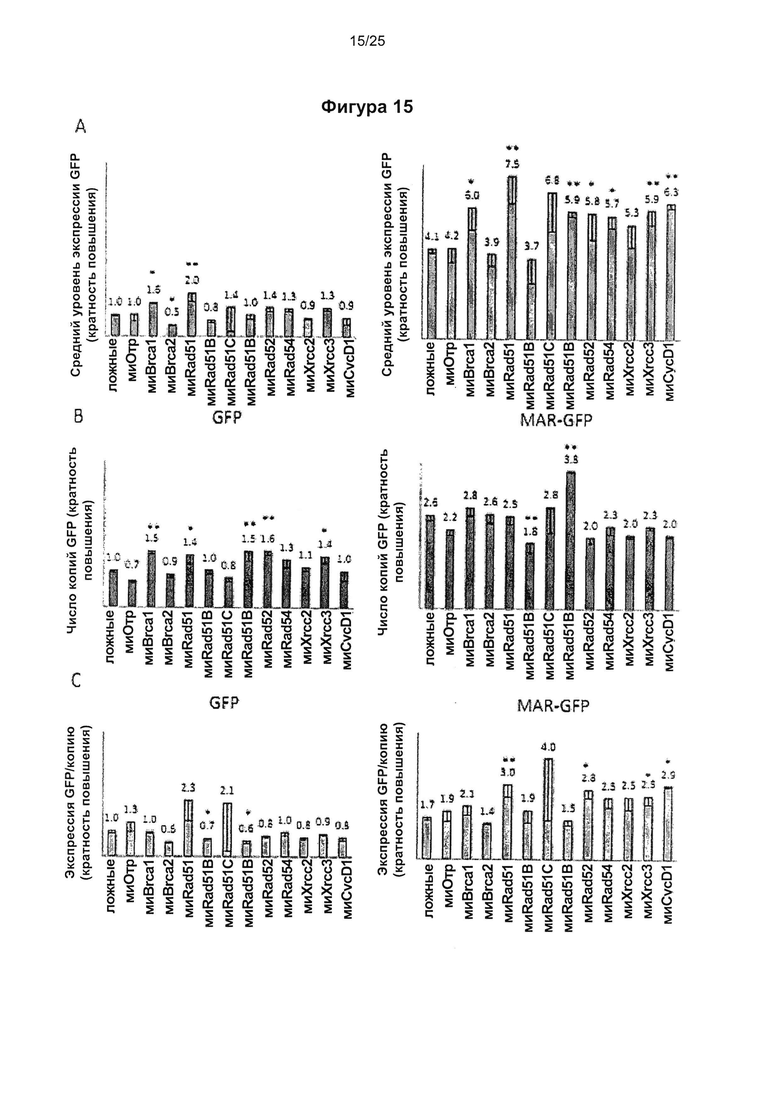
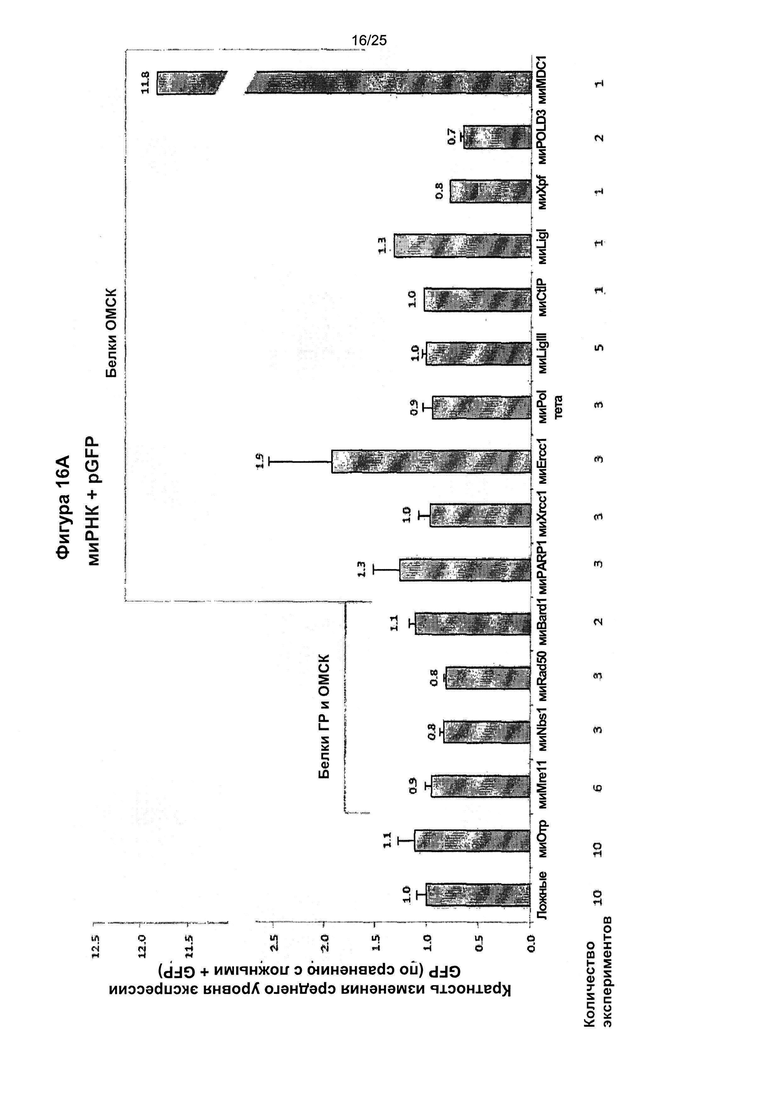
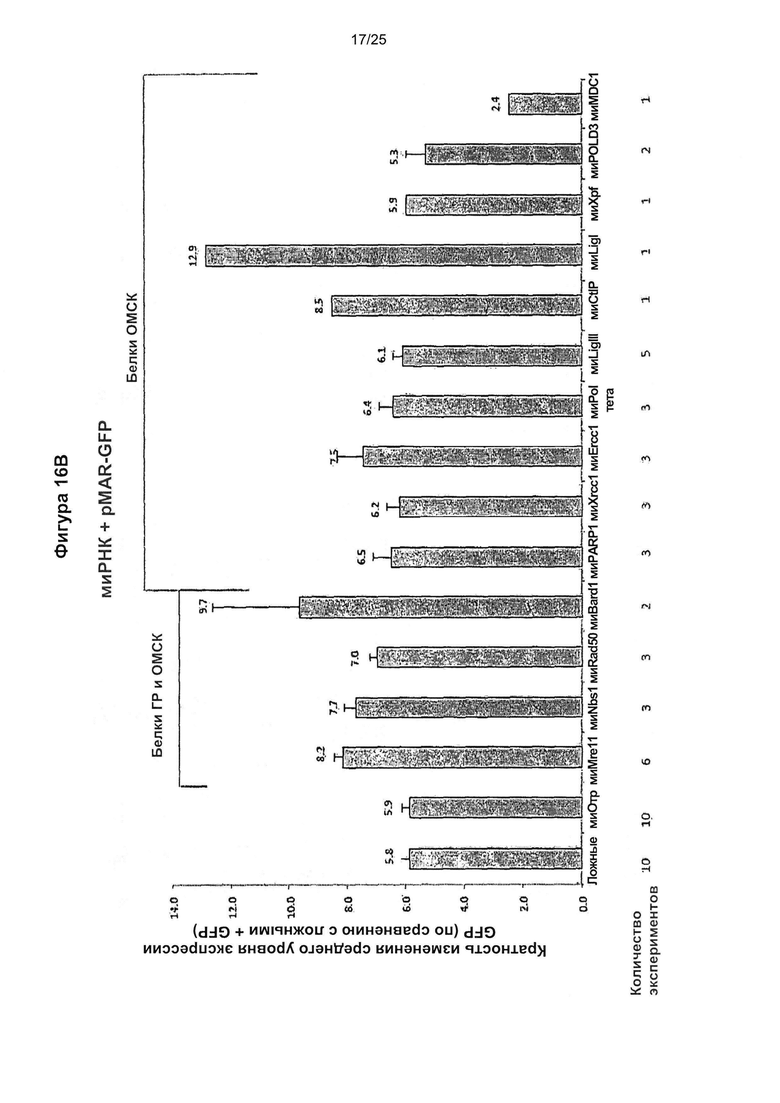
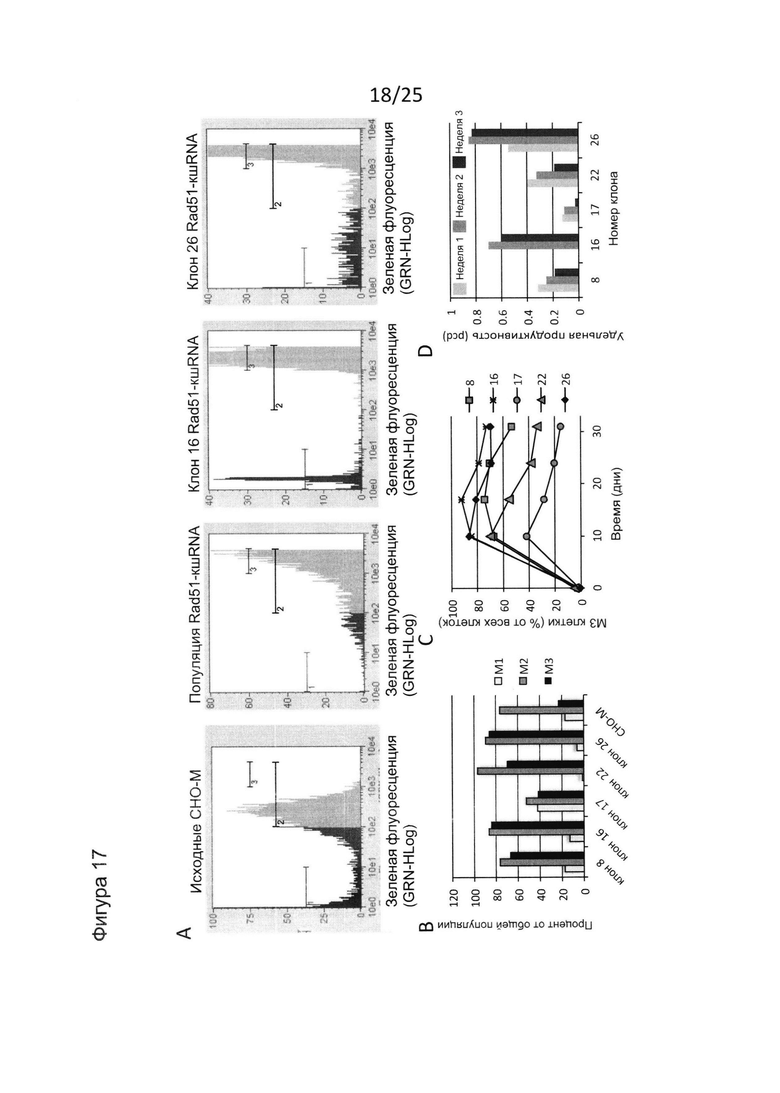
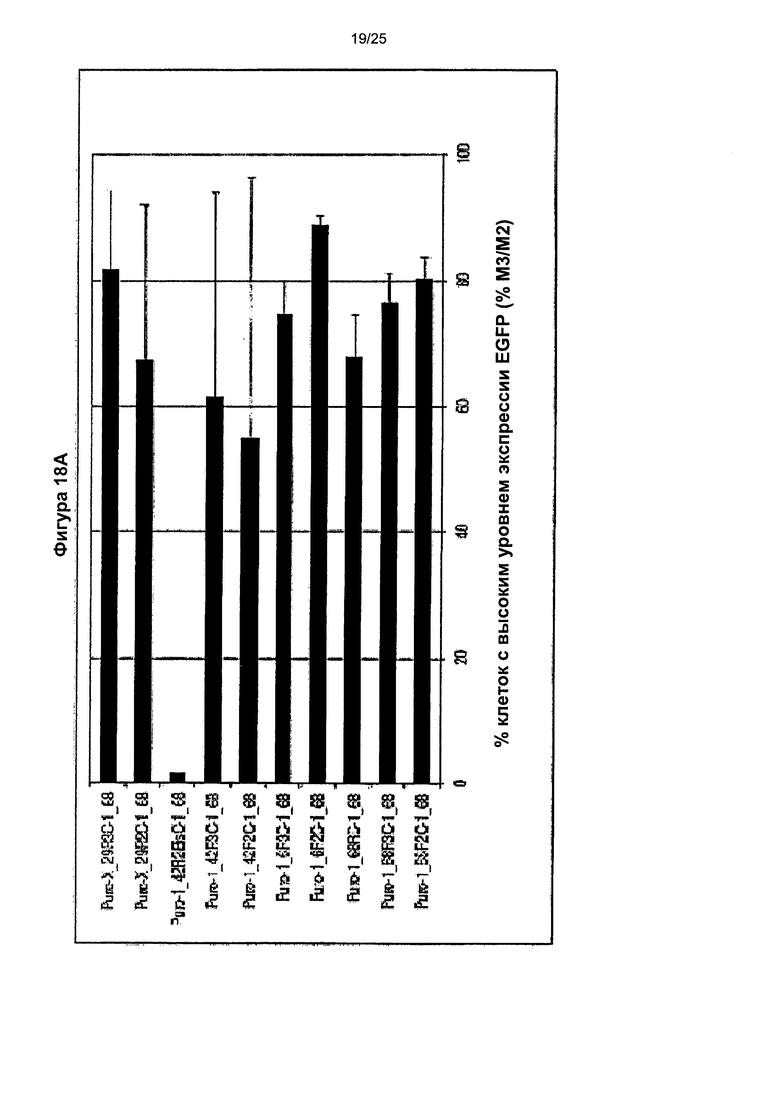
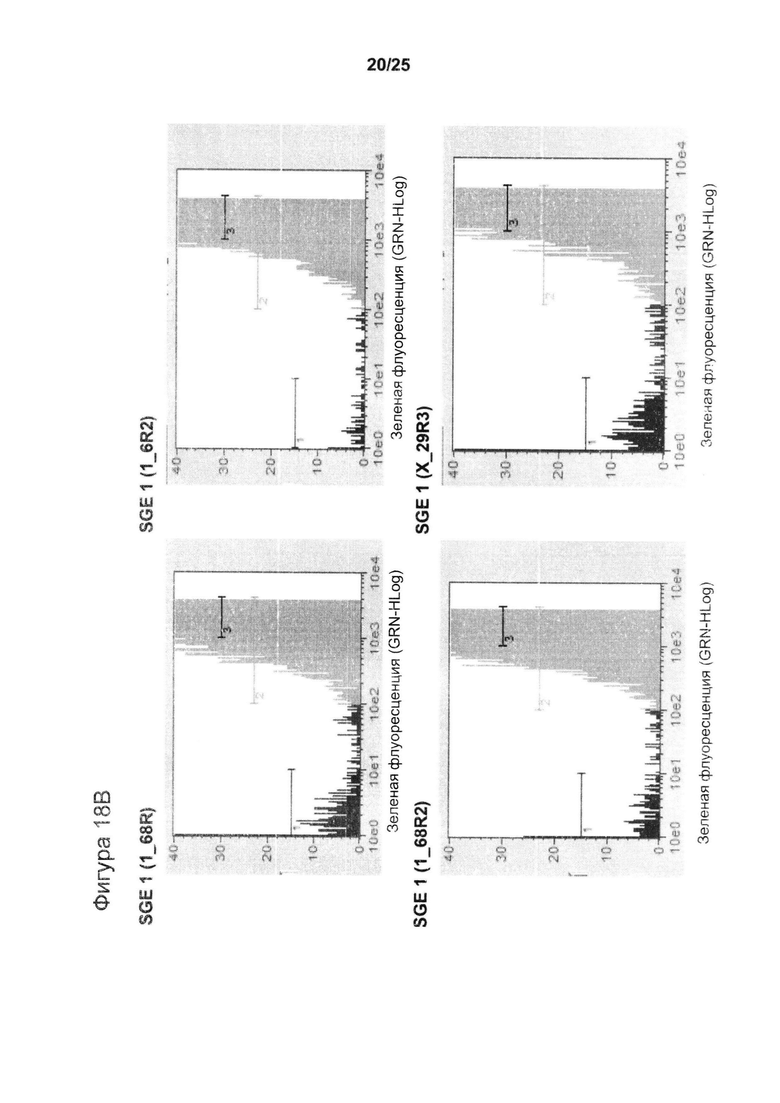
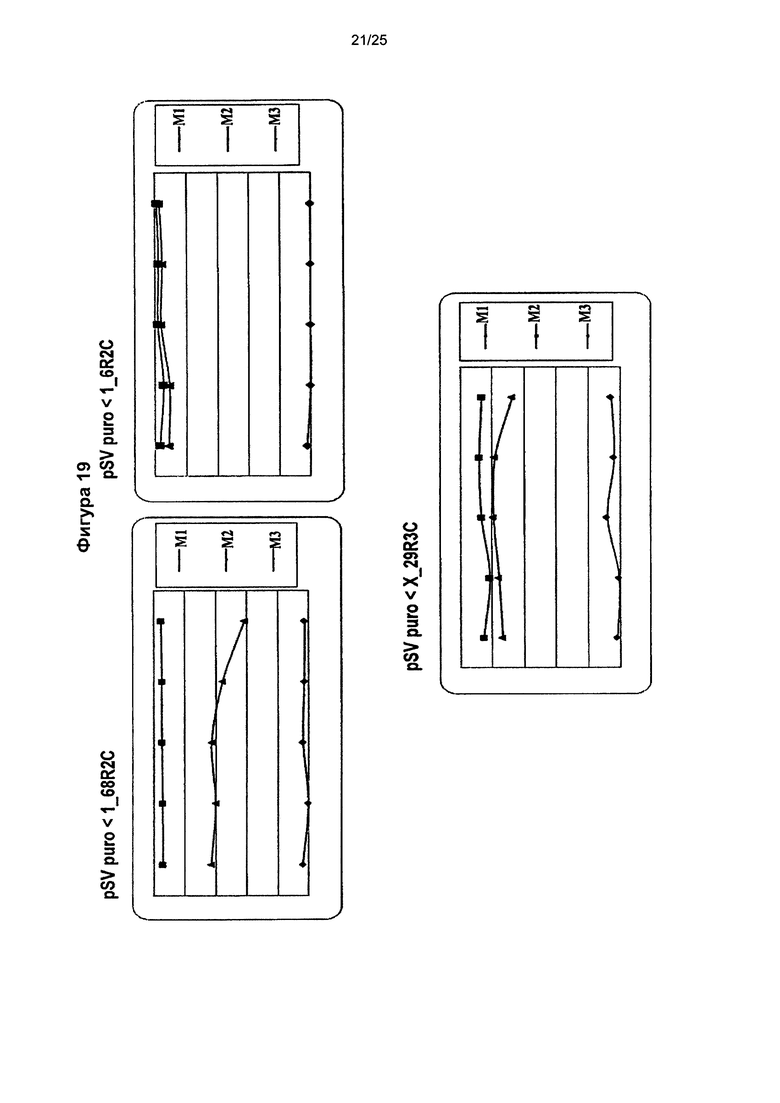
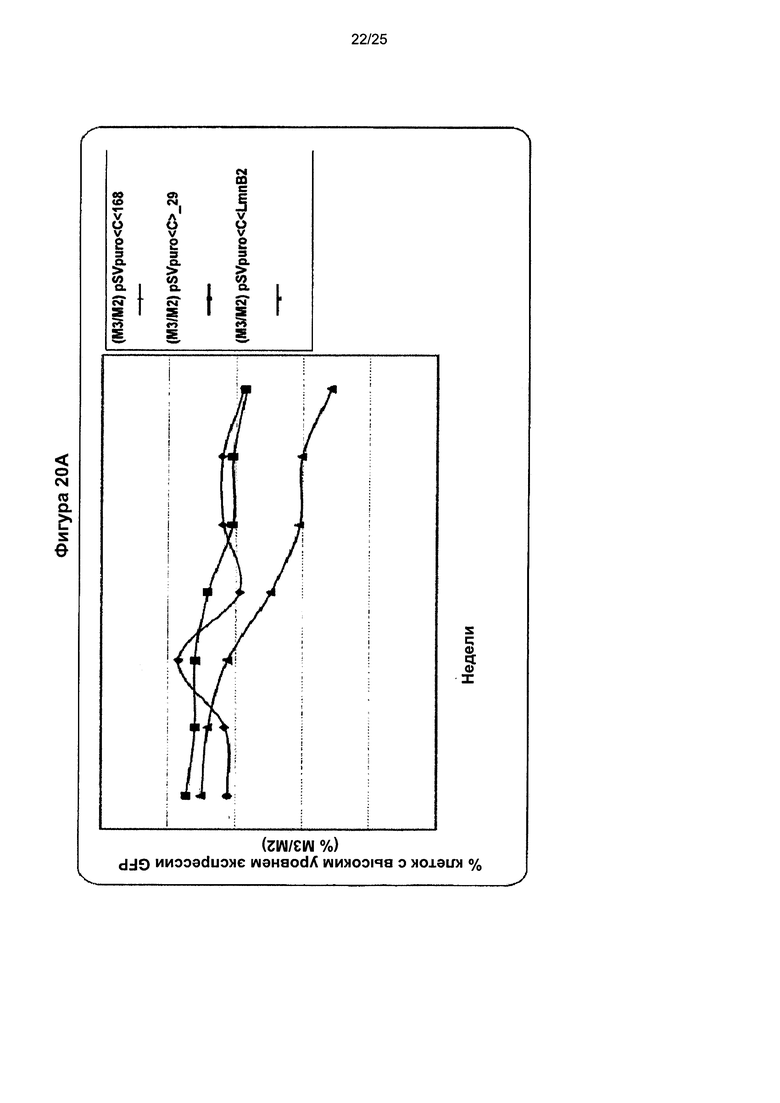
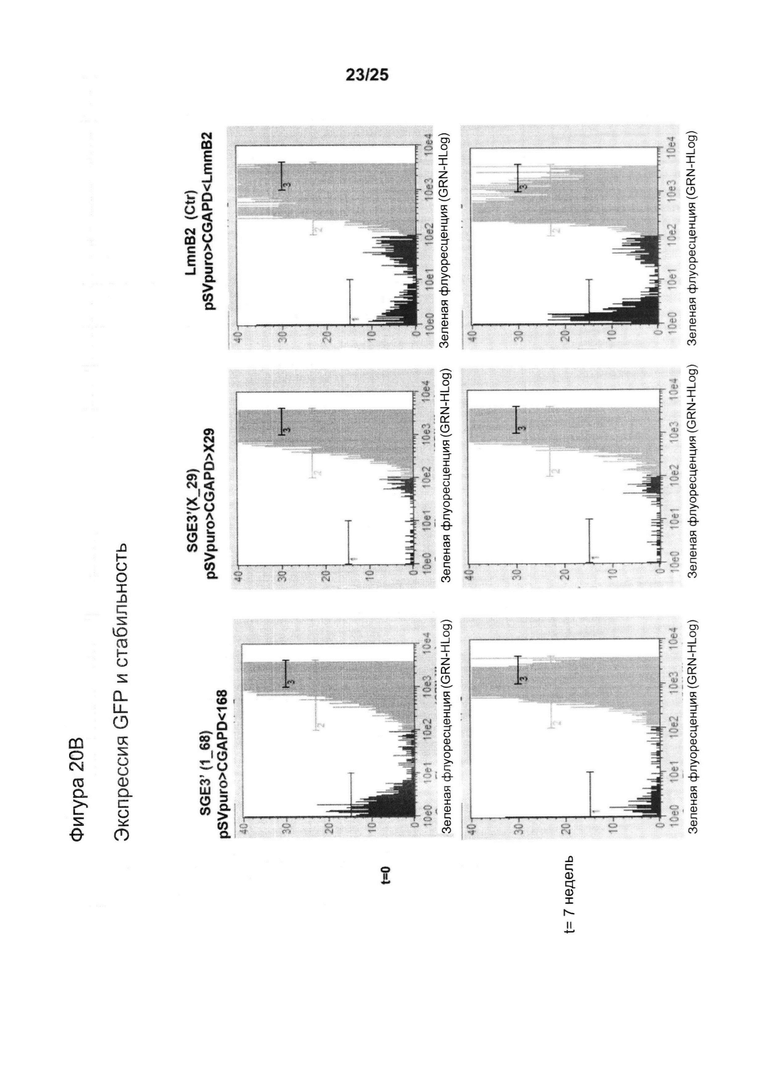
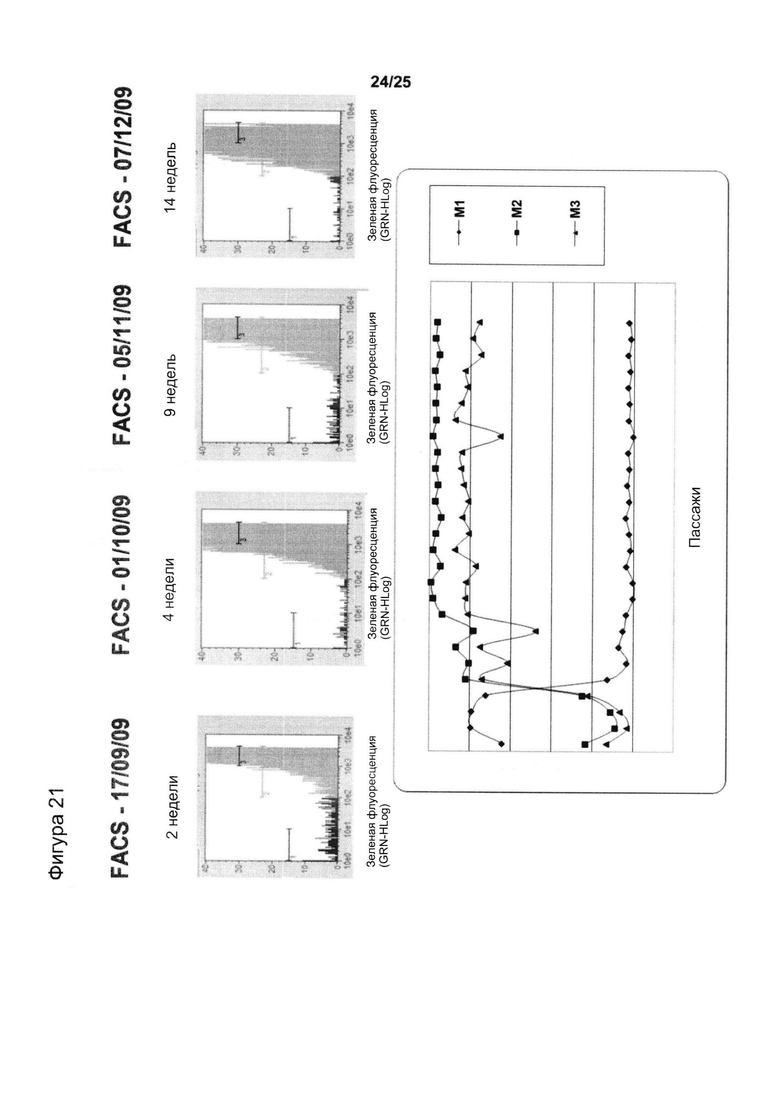
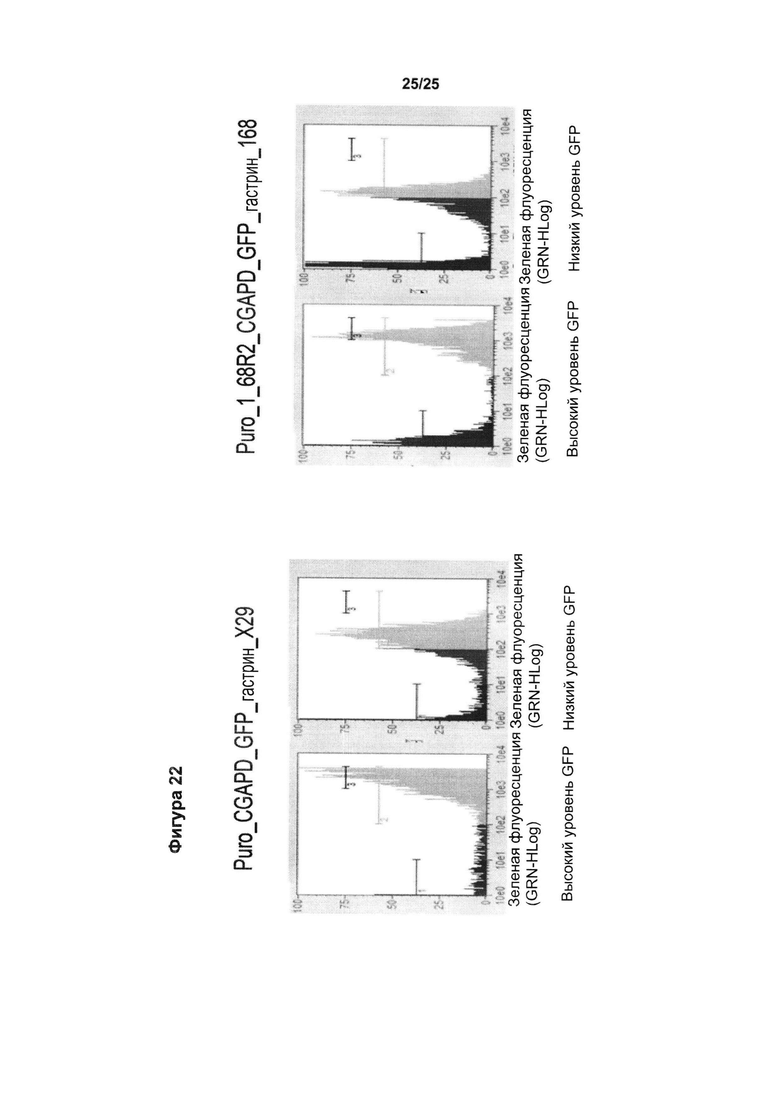
Изобретение относится к области биотехнологии, конкретно к рекомбинантной молекуле нуклеиновой кислоты для увеличения эффективности продукции трансгена, экспрессируемого в клетке млекопитающего. Рекомбинантная молекула нуклеиновой кислоты по настоящему изобретению сконструирована таким образом, что включает 5’- 3’-транспозон-специфичный концевой повтор ITR, нуклеиновую кислоту, кодирующую белок трансгена TEP или РНК TEP, а также дополнена эпигенетическим регуляторным элементом. Кроме того, описаны рекомбинантный экспрессионный вектор, клетка-хозяин, способ экспрессии кодируемого ТЕР, способ трансфекции клетки и набор для осуществления трансфекции клетки. В сравнении с известными аналогами изобретение демонстрирует преимущества в отношении величины и качества экспрессии, включая высокую стабильность экспрессии трансгена и, при необходимости, транспорт экспрессированного продукта наружу из клетки. 11 н. и 53 з.п. ф-лы, 4 табл., 25 ил.
1. Рекомбинантная молекула нуклеиновой кислоты для увеличения эффективности продукции трансгена, экспрессируемого в клетке млекопитающего, содержащая:
(a) 5'- и 3'-транспозон-специфичный инвертированный концевой повтор (ITR),
(b) по меньшей мере одну последовательность нуклеиновой кислоты, кодирующую белок экспрессии/процессинга трансгена (ТЕР) или функциональную РНК ТЕР, расположенную между 5'- и 3'-ITR, и которая находится под контролем промотора, который обеспечивает экспрессию указанного белка экспрессии/процессинга трансгена (ТЕР) или функциональной РНК ТЕР в клетке млекопитающего; и
(c) по меньшей мере один эпигенетический регуляторный элемент, представляющий собой MAR (участок прикрепления к матриксу), который расположен между 5'- и 3'-ITR;
при этом указанный белок ТЕР представляет собой один или более из следующих белков пути секреции белка: hSRP14, hSec61α1, hSec61β, hSec61γ, hSRP54, hSRP9, hSRPRα, hSRPβ и hCANX,
или белок ТЕР соответствует одной или более из следующих последовательностей аминокислот белков пути секреции белка: hSRP14 с последовательностью SEQ ID NO: 13, hSec61α1 с последовательностью SEQ ID NO: 15, hSec61β с последовательностью SEQ ID NO: 17, hSec61γ с последовательностью SEQ ID NO: 19, hSRP54 с последовательностью SEQ ID NO: 21, hSRP9 с последовательностью SEQ ID NO: 23, hSRPRα с последовательностью SEQ ID NO: 25, hSRPβ с последовательностью SEQ ID NO: 27, и hCANX с последовательностью SEQ ID NO: 29 и последовательностям аминокислот, идентичным более чем на 80%, 90%, 95% или 98% указанным последовательностям;
причем указанная функциональная РНК ТЕР внутри указанной клетки включает/состоит из последовательности нуклеиновых кислот, кодирующей функциональную РНК, предпочтительно микроРНК или кшРНК, которая препятствует экспрессии по меньшей мере одного белка пути рекомбинации, такого как, но не ограничиваясь перечисленными: Rad51, Rad51B, Rad51C, Rad51D, Xrcc2, Xrcc3, Rad52, Rad54, Brca1, Brca2, циклин D1, Ercc, MDC1, Bard1, лигаза 1, Mre11; и
при этом указанная клетка млекопитающего представляет собой клетку грызуна.
2. Рекомбинантная молекула нуклеиновой кислоты по п. 1, отличающаяся тем, что уровень экспрессии трансгена в указанной клетке млекопитающего увеличен на по меньшей мере на 10%.
3. Рекомбинантная молекула нуклеиновой кислоты по любому из пп. 1 или 2, отличающаяся тем, что указанный элемент MAR выбран из последовательностей SEQ ID NO: 1 (MAR 1-68), 2 (MAR 1_6), 3 (MAR X_S29), 4 (MAR S4), 5 (MAR лизоцима цыпленка), представляет собой сконструированный, в частности, перестроенный аналог и/или его последовательность по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентична любой из последовательностей SEQ ID NO: с 1 по 5.
4. Рекомбинантная молекула нуклеиновой кислоты по любому из пп. 1-3, отличающаяся тем, что указанная функциональная РНК ТЕР представляет собой микроРНК или кшРНК.
5. Рекомбинантная молекула нуклеиновой кислоты по любому из пп. 1-3, отличающаяся тем, что длина указанной молекулы нуклеиновой кислоты составляет по меньшей мере 5000, 6000, 7000, 8000, 9000 или 10000 п.о.
6. Рекомбинантная молекула нуклеиновой кислоты по любому из пп. 1-3, отличающаяся тем, что 5'- и 3'-ITR представляют собой 5'- и 3'-ITR транспозона Спящая Красавица или предпочтительно транспозона PiggyBac.
7. Рекомбинантная молекула нуклеиновой кислоты по любому из пп. 1-3, отличающаяся тем, что после первой трансфекции клетки млекопитающего указанной рекомбинантной молекулой нуклеиновой кислоты и второй последующей трансфекции дополнительной рекомбинантной молекулой нуклеиновой кислоты, содержащей трансген, встраивание и/или экспрессия трансгена оказываются повышенными в указанной клетке по сравнению с клеткой, не подвергнутой указанной первой трансфекции.
8. Вектор экспрессии для экспрессии ТЕР или функциональной РНК ТЕР, содержащий рекомбинантную молекулу нуклеиновой кислоты по любому из пп. 1-7.
9. Вектор по п. 8, отличающийся тем, что указанный вектор дополнительно содержит единичный элемент MAR, два или более элементов MAR, при этом указанный элемент(ы) расположен между 5'- и 3'-ITR.
10. Вектор по п. 8, отличающийся тем, что указанный вектор содержит два элемента MAR, при этом первый элемент MAR расположен в 3'-5' направлении от ТЕР или функциональной РНК ТЕР, и второй элемент MAR расположен в 5'-3' направлении от ТЕР или функциональной РНК ТЕР, при этом указанный первый элемент MAR включает элемент MAR 1_6 и/или элемент, последовательность которого по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентична последовательности SEQ ID NO: 2, в частности, перестроенные MAR на основе MAR 1-6, а конкретнее, элементы, последовательность которых по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентична SEQ ID NO: 8 (MAR 1_6R2), и второй элемент MAR включает элемент MAR 1-68 и/или элемент, последовательность которого по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентична последовательности SEQ ID NO: 1.
11. Вектор по п. 8, отличающийся тем, что указанный вектор содержит единичный элемент MAR, при этом указанный единичный элемент MAR расположен в 5'-3' направлении от ТЕР или функциональной РНК ТЕР, и при этом единичный элемент MAR представляет собой MAR 1-68, или элемент MAR Х-29, и/или элемент, последовательность которого по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентична последовательности SEQ ID NO: 1 или 3, в частности, перестроенный MAR на основе MAR 1–68 или MAR Х-29, в частности, элемент, последовательность которого по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентична последовательности SEQ ID NO: 6, 7 или 10 (элементы MAR 1_68R, 1_68R2 или X_29R3) или 9, и предпочтительно представляет собой элемент MAR Х-29 и/или элемент, последовательность которого по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентична последовательности SEQ ID NO: 3.
12. Вектор по п. 8, отличающийся тем, что указанный ТЕР или функциональная РНК ТЕР находится под контролем промотора EF1-альфа, и за ним возможно следует поли(А) сигнал BGH.
13. Вектор по п. 8, отличающийся тем, что указанный вектор содержит промотор(ы) и/или энхансер(ы) или продукты их слияния, такие как GAPDH, CGAPDH, SV40p, CMVp, СНО EF1-альфа, СНО Actb и/или СНО Hspa5.
14. Способ экспрессии ТЕР или функциональной РНК ТЕР, включающий:
обеспечение рекомбинантной клетки млекопитающего, содержащей трансген, и вектора по по любому из пп. 8-13;
осуществление трансфекции указанной клетки указанным вектором с обеспечением экспрессии указанного ТЕР или функциональной РНК ТЕР в клетке, причем указанная клетка является клеткой грызуна.
15. Способ по п. 14, отличающийся тем, что ТЕР или функциональная РНК ТЕР, экспрессируемая с помощью указанного вектора, повышает экспрессию трансгена в указанной клетке млекопитающего на по меньшей мере 10%, по меньшей мере 20%, по меньшей мере 30%, по меньшей мере 40%, по меньшей мере 50%, по меньшей мере 60% или по меньшей мере 70%.
16. Способ по п. 14, отличающийся тем, что указанный вектор содержит единичный элемент MAR Х-29 и/или последовательность нуклеиновой кислоты, которая по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентична последовательности SEQ ID NO: 3, и тем, что через более чем 4, 5, 6, 7, 8, 9, 10, 11, 12, 13 или 14 недель культивирования ТЕР или функциональная РНК ТЕР, экспрессируемая с указанного вектора, повышает экспрессию интересующего гена на по меньшей мере 10%, по меньшей мере 20%, по меньшей мере 30%, по меньшей мере 40%, по меньшей мере 50%, по меньшей мере 60% или по меньшей мере 70%.
17. Рекомбинантная клетка млекопитающего для экспрессии трансгена, содержащая не более чем 20, 15, 10 или 5 рекомбинантных молекул нуклеиновых кислот по пп. 2-13 или вектор по п. 15 и по меньшей мере один трансген, причем указанная клетка представляет собой клетку грызуна.
18. Рекомбинантная клетка млекопитающего по п. 17, отличающаяся тем, что указанные рекомбинантные молекула или вектор встроены в геном указанной клетки в виде единичных копий.
19. Рекомбинантная клетка млекопитающего по любому из пп. 17-18, отличающаяся тем, что указанная клетка представляет собой первичную стволовую клетку, клетку хомячка, например, СНО (клетку яичника китайского хомячка).
20. Рекомбинантная клетка млекопитающего для продукции трансгена, содержащая:
а) рекомбинантную молекулу нуклеиновой кислоты по любому из пп. 1-7 или вектор по п. 8; и
б) рекомбинантную молекулу нуклеиновой кислоты, содержащую по меньшей мере один представляющий интерес трансген, который находится под контролем промотора трансгена, причем указанная клетка представляет собой клетку грызуна.
21. Рекомбинантная клетка млекопитающего по п. 20, которая дополнительно содержит элемент MAR.
22. Рекомбинантная клетка млекопитающего по любому из пп. 20-21, отличающаяся тем, что указанная функциональная РНК представляет собой временно трансфицированную миРНК или кшРНК, которая транскрибируется с указанной по меньшей мере одной изолированной последовательности нуклеиновой кислоты, при этом указанная миРНК или указанная процессированная кшРНК представляет собой антисмысловую РНК длиной 20, 21, 22, 23, 24 или 25 пар оснований, которая полностью комплементарна 20, 21, 22, 23, 24 или 25 последовательным нуклеотидам мРНК по меньшей мере одного целевого гена, который является частью рекомбинантного пути НГСК (негомологичного соединения концов), ГР (гомологичной рекомбинации), ОМСК (опосредованного микрогомологией соединения концов) или представляет собой белок репарации ДНК, такой как MDC1 (медиатор контрольных точек повреждения ДНК 1).
23. Рекомбинантная клетка млекопитающего по п. 22, отличающаяся тем, что указанный по меньшей мере один целевой ген, который является частью:
- НГСК, представляет собой 53 ВР1 (белок 1, связывающий опухолевый супрессор р53),
- ГР, представляет собой
Rad51 (белок репарации ДНК RAD51), Rad51B (гомолог 2 белка репарации ДНК RAD51), Rad51C (гомолог 3 белка репарации ДНК RAD51),
Rad51D (гомолог 4 белка репарации ДНК RAD51), Rad52 (белок репарации ДНК RAD52), Rad54 (белок репарации и рекомбинации ДНК RAD54),
Xrcc2 (репарация после облучения рентгеновскими лучами, комплементирующая нарушенную репарацию в клетках китайского хомячка 2),
Xrcc3 (репарация после облучения рентгеновскими лучами, комплементирующая нарушенную репарацию в клетках китайского хомячка 3),
Brca1 (рак молочной железы 1, раннее начало),
Brca2 (рак молочной железы 2, раннее начало),
Bard1 (связанный с BRCA1 домен RING 1),
- ОМСК, представляет собой
Ercc1 (эксцизионная репарация, кросс-комплементирующая нарушенную репарацию у грызунов, группа комплементации 1),
Mre11 (мейотическая рекомбинация 11)
лигазу 1 (ДНК-лигаза 1),
и/или
представляет собой белок репарации ДНК MDC1.
24. Рекомбинантная клетка млекопитающего по любому из пп. 20, 21 или 23, отличающаяся тем, что указанный по меньшей мере один трансген экспрессирует терапевтический белок, такой как иммуноглобулин, гормон, такой как эритропоэтин, или фактор роста, и при этом необязательно встраивание и/или экспрессия трансгена повышены в рекомбинантной клетке млекопитающего по сравнению с клеткой, не содержащей указанную рекомбинантную молекулу(ы) нуклеиновой кислоты.
25. Рекомбинантная клетка млекопитающего по любому из пп. 20, 21 или 23, отличающаяся тем, что указанная клетка содержит по меньшей мере две функциональные РНК ТЕР, при этом одна или обе РНК ТЕР представляют собой временно трансфицированные миРНК или экспрессируются с указанной изолированной последовательности(ей) нуклеиновой кислоты, кодирующей функциональную РНК ТЕР.
26. Рекомбинантная клетка млекопитающего по любому из пп. 20, 21 или 23, отличающаяся тем, что указанная рекомбинантная клетка млекопитающего содержит элемент MAR.
27. Рекомбинантная молекула нуклеиновой кислоты для увеличения эффективности продукции трансгена, экспрессируемого в клетке млекопитающего, содержащая:
(а) 5'- и 3'-транспозон-специфичный инвертированный концевой повтор (ITR),
(b) по меньшей мере одну последовательность нуклеиновой кислоты, кодирующую белок экспрессии/процессинга трансгена (ТЕР) или функциональную РНК ТЕР, расположенную между 5'- и 3'-ITR, и которая находится под контролем промотора, который обеспечивает экспрессию указанного белка экспрессии/процессинга трансгена (ТЕР) или функциональной РНК ТЕР в клетке млекопитающего; и
(c) по меньшей мере один эпигенетический регуляторный элемент, представляющий собой MAR (участок прикрепления к матриксу), который расположен между 5'- и 3'-ITR,
(d) по меньшей мере один трансген, также расположенный между 5'- и 3'-ITR, который находится под контролем промотора трансгена;
при этом указанный белок ТЕР представляет собой один или более из следующих белков пути секреции белка: hSRP14, hSec61α1, hSec61β, hSec61γ, hSRP54, hSRP9, hSRPRα, hSRPβ и hCANX, или белок ТЕР соответствует одной или более из следующих последовательностей аминокислот белков пути секреции белка: hSRP14 с последовательностью SEQ ID NO: 13, hSec61α1 с последовательностью SEQ ID NO: 15, hSec61β с последовательностью SEQ ID NO: 17, hSec61γ с последовательностью SEQ ID NO: 19, hSRP54 с последовательностью SEQ ID NO: 21, hSRP9 с последовательностью SEQ ID NO: 23, hSRPRα с последовательностью SEQ ID NO: 25, hSRPβ с последовательностью SEQ ID NO: 27, и hCANX с последовательностью SEQ ID NO: 29 и последовательностям аминокислот, идентичным более чем на 80%, 90%, 95% или 98% указанным последовательностям;
причем указанная функциональная РНК ТЕР внутри указанной клетки включает/состоит из последовательности нуклеиновых кислот, кодирующей функциональную РНК, предпочтительно микроРНК или кшРНК, которая препятствует экспрессии по меньшей мере одного белка пути рекомбинации, такого как, но не ограничиваясь перечисленными: Rad51, Rad51B, Rad51C, Rad51D, Xrcc2, Xrcc3, Rad52, Rad54, Brca1, Brca2, циклин D1, Ercc, MDC1, Bard1, лигаза 1 или Mre11; и
при этом указанная клетка млекопитающего представляет собой клетку грызуна.
28. Рекомбинантная молекула нуклеиновой кислоты по п. 27, отличающаяся тем, что указанный элемент MAR выбран из последовательностей SEQ ID NO: 1 (MAR 1-68), 2 (MAR 1_6), 3 (MAR X_S29), 4 (MAR S4), 5 (MAR лизоцима цыпленка), представляет собой сконструированный, в частности, перестроенный аналог и/или его последовательность по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентична любой из последовательностей SEQ ID NO: с 1 по 5.
29. Рекомбинантная молекула нуклеиновой кислоты по любому из пп. 27-28, отличающаяся тем, что указанная функциональная РНК ТЕР представляет собой микроРНК или кшРНК.
30. Рекомбинантная молекула нуклеиновой кислоты по любому из пп. 27-28, отличающаяся тем, что длина указанной молекулы нуклеиновой кислоты составляет по меньшей мере 5000, 6000, 7000, 8000, 90000 или 10000 п.о.
31. Рекомбинантная молекула нуклеиновой кислоты по любому из пп. 27-28, отличающаяся тем, что 5'- и 3'-ITR представляют собой 5'- и 3'-ITR транспозона Спящая Красавица или предпочтительно транспозона PiggyBac.
32. Рекомбинантная молекула нуклеиновой кислоты по любому из пп. 27-28, отличающаяся тем, что после первой трансфекции клетки млекопитающего указанной рекомбинантной молекулой нуклеиновой кислоты и второй последующей трансфекции дополнительной рекомбинантной молекулой нуклеиновой кислоты, содержащей трансген, встраивание и/или экспрессия трансгена оказываются повышенными в указанной клетке по сравнению с клеткой, не подвергнутой указанной первой трансфекции.
33. Рекомбинантная молекула кислоты по п. 27, отличающаяся тем, что указанный элемент MAR расположен между 5'- и 3'-ITR и что необязательно трансген, такой как ген устойчивости к антибиотику или ген, кодирующий иммуноглобулин, возможно находящийся под контролем дополнительного промотора, расположен между 5'- и 3'-ITR, например, между 5'-ITR и MAR.
34. Способ трансфекции клетки млекопитающего, включающий:
трансфекцию указанных клеток млекопитающих
(i) по меньшей мере одной, по меньшей мере двумя, по меньшей мере тремя или по меньшей мере четырьмя рекомбинантными молекулами нуклеиновой кислоты по любому из пп. 27–33, или
(ii) вектором по п. 8 и/или по меньшей мере одной, по меньшей мере двумя, по меньшей мере тремя или по меньшей мере четырьмя изолированными функциональными РНК ТЕР, экспрессируемыми рекомбинантной молекулой нуклеиновой кислоты по любому из пп. 27-33 или вектором по п. 8, и
(iii) по меньшей мере одним трансгеном, который находится под контролем промотора трансгена,
причем указанная клетка представляет собой клетку грызуна.
35. Способ по п. 34, отличающийся тем, что трансфекцию указанной рекомбинантной молекулы нуклеиновой кислоты по пп. 27-33 или вектора по п. 8 осуществляют в качестве первой трансфекции, и/или трансфекцию указанного трансгена, который необязательно является частью рекомбинантной молекулы нуклеиновой кислоты, возможно осуществляют в качестве второй, последующей трансфекции, возможно вместе с изолированной нуклеиновой кислотой или мРНК, экспрессирующей транспозазу, которая распознает указанные 5'- и 3'-ITR.
36. Способ по п. 34, отличающийся тем, что указанные клетки представляют собой клетки хомячка.
37. Способ по п. 34, отличающийся тем, что указанную клетку трансфицируют более чем одной рекомбинантной молекулой нуклеиновых кислот, включая по меньшей мере две, по меньшей мере три или по меньшей мере четыре из указанных рекомбинантных молекул нуклеиновых кислот, кодирующих один, два или три из указанных белков ТЕР.
38. Способ по п. 34 или 35, отличающийся тем, что каждая из указанных молекул нуклеиновых кислот является частью вектора, и тем, что осуществляют котрансфекцию указанными векторами.
39. Способ по п. 38, отличающийся тем, что указанная котрансфекция, предпочтительно котрансфекция рекомбинантными молекулами нуклеиновых кислот, кодирующими белки SRP14, SRP9 и SRP54, повышает встраивание и/или экспрессию трансгена в указанной клетке по сравнению с клеткой, не подвергнутой такой котрансфекции.
40. Способ по п. 34, отличающийся тем, что множество указанных клеток млекопитающих, которые стабильно экспрессируют указанный белок ТЕР или функциональную РНК ТЕР, получают, чтобы получить рекомбинантные клетки млекопитающих, и при этом указанное множество рекомбинантных клеток млекопитающих не зависит от присутствия указанного элемента MAR.
41. Способ по п. 34, отличающийся тем, что указанные клетки млекопитающих трансфицируют второй раз.
42. Способ по п. 34, отличающийся тем, что по меньшей мере 30%, 40% или 45% указанных клеток млекопитающих становятся рекомбинантными клетками млекопитающих и экспрессируют указанный трансген.
43. Способ по п. 34, отличающийся тем, что указанная клетка млекопитающего трансфицирована указанной по меньшей мере одной изолированной функциональной РНК ТЕР и вектором, содержащим указанный по меньшей мере один трансген и необязательно 3'-ITR и 5'-ITR, фланкирующие указанный по меньшей мере один трансген, возможно вместе с изолированной нуклеиновой кислотой или мРНК, экспрессирующей транспозазу, которая распознает указанные 5'- и 3'-ITR.
44. Способ по п. 34, отличающийся тем, что указанный трансген представляет собой терапевтический белок, такой как иммуноглобулин, гормон, цитокин или фактор роста.
45. Способ по п. 34, отличающийся тем, что указанная рекомбинантная молекула нуклеиновой кислоты необязательно содержит маркер отбора и что указанный по меньшей мере один трансген экспрессируется
(a) без отбора по указанному маркеру, или
(b) с отбором по указанному маркеру, например, путем добавления селекционного агента в культуральную среду, и
(c) в отсутствие транспозазы, или
(d) в присутствии транспозазы.
46. Способ по п. 34, отличающийся тем, что встраивание и/или экспрессия трансгена повышены в такой клетке по сравнению с клеткой, не трансфицированной указанными изолированными молекулами нуклеиновых кислот и/или по меньшей мере одной из указанных изолированных функциональных РНК ТЕР.
47. Набор для осуществления трансфекции клетки, в одном контейнере содержащий по меньшей мере один вектор, содержащий любую из указанных рекомбинантных молекул нуклеиновых кислот по пп. 1-7 или 27-33, или вектор по п. 8, и в дополнительном контейнере содержащий инструкцию по применению указанного вектора или векторов, причем указанная клетка представляет собой клетку грызуна.
48. Набор по п. 47, отличающийся тем, что указанный набор дополнительно к указанным двум контейнерам содержит в еще одном дополнительном контейнере вектор, кодирующий подходящую транспозазу.
49. Набор по п. 47, отличающийся тем, что более чем один вектор предусмотрен в одном или более контейнерах, и что указанные белки ТЕР представляют собой по меньшей мере два из следующих белков: шаперон, SRP14, SRP9 или SRP54.
50. Применение рекомбинантной молекулы нуклеиновой кислоты по любому из пп. 1-7, 27-33 для повышения встраивания трансгена в геном клетки хозяина и/или экспрессии трансгена, причем указанная клетка представляет собой клетку грызуна.
51. Применение по п. 50, отличающееся тем, что указанная рекомбинантная молекула нуклеиновой кислоты является частью вектора экспрессии.
52. Вектор экспрессии, содержащий рекомбинантную молекулу нуклеиновой кислоты по любому из пп. 1-7, и дополнительно содержащий:
(a) трансген, который фланкирован в 3'-5' направлении промотором и в 5'-3' направлении сигналом полиаденилирования,
(b) единичный элемент MAR в 5'-3' направлении от сигнала полиаденилирования, или
(c) первый элемент MAR в 3'-5' направлении от интересующего трансгена и второй элемент MAR в 5'-3' направлении от места встраивания указанного трансгена.
53. Вектор экспрессии по п. 52, отличающийся тем, что указанный единичный или первый и второй элементы MAR выбраны из элементов MAR 1_68, 1_6, 1_6R2, 1_68R, 1_68R2, X_29R3, или X_29 или элементов, последовательности которых по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентичны последовательностям SEQ ID NO: 1, 2, 3, 6, 7, 8, 9 или 10.
54. Вектор экспрессии по п. 52, отличающийся тем, что указанный единичный элемент или первый и второй элементы MAR выбран/выбраны из перестроенных элементов MAR 1_6R2, 1_68R, 1_68R2 или X_29R3 или элементов, последовательности которых по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентичны последовательностям SEQ ID NO: 6, 7, 8 или 10, при этом необязательно указанный элемент(ы) MAR повышает экспрессию интересующего трансгена на по меньшей мере 10%, по меньшей мере 20%, по меньшей мере 30%, по меньшей мере 40%, по меньшей мере 50%, по меньшей мере 60% или по меньшей мере 70% по сравнению с их неперестроенными аналогами.
55. Вектор экспрессии по пп. 52-54, отличающийся тем, что указанный промотор представляет собой промотор EF1-альфа и указанный сигнал полиаденилирования представляет собой поли(А) сигнал BGH.
56. Вектор экспрессии по пп. 52-54, отличающийся тем, что указанный вектор содержит промотор(ы), и/или энхансер(ы), или продукты их слияния, такие как GAPDH, CGAPDH, CSV40p, CMVp, СНО Ef1-альфа, СНО Actb или СНО Hspa5.
57. Вектор экспрессии по п. 56, отличающийся тем, что указанный промотор представляет собой промотор GAPDH и содержит энхансер CMV.
58. Вектор экспрессии по пп. 52-54 или 57, отличающийся тем, что указанный один или первый и второй MAR, энхансер, промотор, интересующий трансген и сигнал полиаденилирования расположены между 5'- и 3'-ITR.
59. Вектор экспрессии по пп. 52-54 или 57, содержащий
(a) единичный элемент MAR в 5'-3' направлении от сигнала полиаденилирования, при этом указанный единичный элемент MAR предпочтительно представляет собой элемент MAR 1-68 или MARX-29 и/или элемент, последовательность которого по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентична последовательности SEQ ID NO: 1 или 3, в частности, перестроенный MAR на основе MAR 1-68 или MAR Х-29, в частности, элемент, последовательность которого по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентична последовательностям SEQ ID NO: 6, 7 или 10 (элементы MAR 1_68R, 1_68R2 или X_29R3) или SEQ ID NO: 9, и предпочтительно представляет собой элемент MAR Х-29, и/или его последовательность по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентична последовательности SEQ ID NO: 3, или
(b) первый элемент MAR в 3'-5' направлении от интересующего трансгена и второй элемент MAR в 5'-3' направлении от указанного интересующего трансгена, при этом указанный первый элемент MAR, предпочтительно включает элемент 1_6, и/или его последовательность по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентична последовательности SEQ ID NO: 2, в частности, идентична перестроенным MAR на основе MAR 1-6, в частности элементам, последовательности которых по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентичны последовательности SEQ ID NO: 8 (MAR 1_6R2), и второй элемент MAR, который предпочтительно включает элемент MAR 1-68, и/или последовательность которого по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентична последовательности SEQ ID NO: 1.
60. Вектор экспрессии по п. 59, содержащий единичный элемент MAR, отличающийся тем, что указанный единичный элемент MAR расположен в 5'-3' направлении от сайта полиаденилирования и представляет собой MAR 1-68 или MAR Х-29, и/или его последовательность по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентична последовательности SEQ ID NO: 1 или 3, в частности, идентична перестроенным MAR на основе MAR 1-68 или MAR Х-29, в частности, элементам, последовательность которых по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентична последовательностям SEQ ID NO: 6, 7 или 10 (элементы MAR 1_68R, 1_68R2 или X_29R3) или 9, и предпочтительно представляет собой элемент, полученый из MAR Х-29, и/или элемент, последовательность которого по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентична последовательности SEQ ID NO: 3.
61. Вектор экспрессии по п. 59, содержащий первый элемент MAR в 3'-5' направлении от трансгена и второй элемент MAR в 5'-3' направлении от указанного интересующего трансгена, отличающийся тем, что указанный первый элемент MAR включает элемент MAR 1_6, и/или его последовательность по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентична последовательности SEQ ID NO: 2, и указанный второй элемент MAR включает элемент MAR 1_68 и/или его последовательность по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентична последовательности SEQ ID NO: 1.
62. Способ экспрессии трансгена, включающий:
обеспечение рекомбинантной клетки млекопитающего, содержащей один из векторов по пп. 52-61, содержащих указанный трансген, и экспрессирующей указанный трансген, причем указанная клетка представляет собой клетку грызуна.
63. Способ по п. 62, отличающийся тем, что указанный элемент(ы) MAR повышает экспрессию указанного трансгена предпочтительно на по меньшей мере 10%, по меньшей мере 20%, по меньшей мере 30%, по меньшей мере 40%, по меньшей мере 50%, по меньшей мере 60% или по меньшей мере 70%.
64. Способ по п. 62, отличающийся тем, что указанный вектор содержит единичный элемент MARX_29 и/или нуклеиновую кислоту, последовательность которой по меньшей мере на 80%, 90%, 95%, 98%, 99% или 100% идентична последовательности SEQ ID NO: 3, и что через более чем 4, 5, 6, 7, 8, 9, 10, 11, 12, 13 или 14 недель культивирования указанный элемент MAR повышает экспрессию интересующего трансгена на по меньшей мере 10%, по меньшей мере 20%, по меньшей мере 30%, по меньшей мере 40%, по меньшей мере 50%, по меньшей мере 60% или по меньшей мере 70%.
| US 2004235011 A1, 25.11.2004 | |||
| EP 0743365 A2, 20.11.1996 | |||
| WO 2011033375 A2, 24.03.2011 | |||
| WO 2005010046 A1, 03.02.2005 | |||
| MATASCI M | |||
| et al., CHO cell lines generated by PiggyBac transposition, BMC PROCEEDINGS, 2011, v | |||
| Кипятильник для воды | 1921 |
|
SU5A1 |
| Способ очистки нефти и нефтяных продуктов и уничтожения их флюоресценции | 1921 |
|
SU31A1 |
| АДЕНОВИРУСНЫЙ/АЛЬФА-ВИРУСНЫЙ ГИБРИДНЫЙ ВЕКТОР ДЛЯ ЭФФЕКТИВНОГО ВВЕДЕНИЯ И ЭКСПРЕССИИ ТЕРАПЕВТИЧЕСКИХ ГЕНОВ В ОПУХОЛЕВЫЕ КЛЕТКИ | 2005 |
|
RU2394104C2 |
Авторы
Даты
2020-01-17—Публикация
2014-02-01—Подача