Перекрестная ссылка на связанные приложения
[0001] Данная заявка испрашивает приоритет по отношению к патентной заявке США № 62/660023, поданной 19 апреля 2018 г., раскрытие которой включено в данный документ посредством ссылки в полном объеме.
ЗАЯВЛЕНИЕ ОТНОСИТЕЛЬНО ПРАВ НА ИЗОБРЕТЕНИЯ, СОЗДАННЫЕ НА ОСНОВЕ ФЕДЕРАЛЬНО СПОНСИРОВАННОЙ НАУЧНО-ИССЛЕДОВАТЕЛЬСКОЙ РАБОТЫ
[0002] Данное изобретение создано при поддержке Правительства США согласно гранту № HR0011-17-2-0043, присужденному Министерством обороны, Агентством перспективных исследовательских проектов Министерства обороны США, и согласно гранту № R01 DA036858, присужденному Национальным институтом здравоохранения. Правительство США имеет определенные права на данное изобретение.
УРОВЕНЬ ТЕХНИКИ
[0003] Несмотря на то, что редактирование генома считается многообещающим терапевтическим подходом к лечению заболеваний, оно сопряжено с неотъемлемыми рисками из-за потенциальной генотоксичности, связанной с двухцепочечными разрывами (ДЦР). Кроме того, редактирование генома часто связано с эффектом «все или ничего» для целевого гена (т.е. оно вызывает полный нокаут). В отличие от этого целенаправленная инженерия эпигенома не несет риска генотоксичности, вызванной ДЦР; кроме того, она дает возможность создать более дифференцированный эффект на экспрессию генов и, таким образом, обеспечить функционирование от полного сайленсинга до менее выраженного эффекта.
[0004] В данном документе представлены решения для этих и других потребностей в данной области техники.
КРАТКОЕ ОПИСАНИЕ СУЩНОСТИ ИЗОБРЕТЕНИЯ
[0005] В одном аспекте данного изобретения представлен слитый белок, включающий в себя фермент нуклеазо-дефицитную РНК-направляемую эндонуклеазу ДНК, домен, связанный с боксом  (домен KRAB), и домен метилтрансферазы ДНК. В одном аспекте данного изобретения представлен слитый белок любой из последовательностей SEQ ID NO: 1-15.
(домен KRAB), и домен метилтрансферазы ДНК. В одном аспекте данного изобретения представлен слитый белок любой из последовательностей SEQ ID NO: 1-15.
[0006] В одном аспекте данного изобретения представлена последовательность нуклеиновой кислоты, кодирующая слитый белок, как описано в данном документе, включительно с его вариантами реализации и аспектами.
[0007] В одном аспекте данного изобретения представлен комплекс, включающий в себя слитый белок, как описано в данном документе, включительно с его вариантами реализации и аспектами, и полинуклеотид, включающий в себя (1) нацеленную на ДНК последовательность, которая комплементарна целевой полинуклеотидной последовательности, и (2) связывающую последовательность для фермента нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК, при этом фермент нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК связан с полинуклеотидом через связывающую последовательность.
[0008] В одном аспекте данного изобретения представлен вектор, включающий в себя последовательность нуклеиновой кислоты слитого белка, как описано в данном документе, включительно с его вариантами реализации и аспектами.
[0009] В одном аспекте данного изобретения представлена клетка, включающая в себя слитый белок, как описано в данном документе, включительно с его вариантами реализации и аспектами, нуклеиновую кислоту, как описано в данном документе, включительно с ее вариантами реализации и аспектами, комплекс, как описано в данном документе, включительно с его вариантами реализации и аспектами, или вектор, как описано в данном документе, включительно с его вариантами реализации и аспектами.
[0010] В одном аспекте данного изобретения представлен способ сайленсинга последовательности целевой нуклеиновой кислоты в клетке, включая доставку первого полинуклеотида, кодирующего слитый белок, как описано в данном документе, включительно с его вариантами реализации и аспектами, в клетку, содержащую целевую нуклеиновую кислоту, и доставку в клетку второго полинуклеотида, который включает в себя (i) нацеленную на ДНК последовательность, которая комплементарна последовательности целевой нуклеиновой кислоты, и (ii) связывающую последовательность для фермента нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК. Без намерения быть связанными какой-либо теорией, считается, что указанный слитый белок вызывает сайленсинг последовательности целевой нуклеиновой кислоты в клетке путем метилирования хроматина, содержащего последовательность целевой нуклеиновой кислоты, и (или) путем введения репрессивных хроматиновых маркеров в хроматин, содержащий последовательность целевой нуклеиновой кислоты. Таким образом, в аспектах данного изобретения указанный слитый белок вызывает сайленсинг последовательности целевой нуклеиновой кислоты в клетке путем метилирования хроматина, содержащего последовательность целевой нуклеиновой кислоты, и (или) путем введения репрессивных хроматиновых маркеров в хроматин, содержащий последовательность целевой нуклеиновой кислоты.
[0011] В одном аспекте данного изобретения представлен способ сайленсинга последовательности целевой нуклеиновой кислоты в клетке, включая доставку комплекса, как описано в данном документе, включительно с его вариантами реализации и аспектами, в клетку, содержащую целевую нуклеиновую кислоту, при этом указанный комплекс вызывает сайленсинг последовательности целевой нуклеиновой кислоты в клетке. Без намерения быть связанными какой-либо теорией, считается, что указанный комплекс вызывает сайленсинг последовательности целевой нуклеиновой кислоты в клетке путем метилирования хроматина, содержащего последовательность целевой нуклеиновой кислоты, и (или) путем введения репрессивных хроматиновых маркеров в хроматин, содержащий последовательность целевой нуклеиновой кислоты. Таким образом, в аспектах данного изобретения указанный комплекс вызывает сайленсинг последовательности целевой нуклеиновой кислоты в клетке путем метилирования хроматина, содержащего последовательность целевой нуклеиновой кислоты, и (или) путем введения репрессивных хроматиновых маркеров в хроматин, содержащий последовательность целевой нуклеиновой кислоты.
КРАТКОЕ ОПИСАНИЕ ГРАФИЧЕСКИХ МАТЕРИАЛОВ
[0012] На Фиг. 1A-1F описано конструирование белка «все в одном» для длительного генного сайленсинга. На Фиг. 1A представлено схематическое изображение белка «все в одном» (SEQ ID NO: 1) согласно данному изобретению, который содержит домен KRAB, слитый с -N-концом dCas9 (SEQ ID NO: 23), отделенный с помощью линкера GGSGGGS (SEQ ID NO: 17), и Dnmt3A - Dnmt3L на С-конце dCas9 (отделенный с помощью линкера EASGSGRASPGIPGSTR (SEQ ID NO: 19)). На Фиг. 1В представлены схематические изображения слитых с dCas9 эпигенетических модуляторов, протестированных на постоянный генный сайленсинг. Белок dCas9 - KRAB адаптирован из Gilbert et al., Cell 2013 для применения с технологией интерференции CRISPR (CRISPRi). Слитый белок dCas9 - Dnmt3A - Dnmt3L адаптирован из Stepper et al., Nucleic Acids Research, 2016. Авторы данного изобретения сконструировали новый белок «все в одном», который объединяет домен KRAB (SEQ ID NO: 16), dCas9 (D10A, H208A), Dnmt3A - Dnmt3L (SEQ ID NO: 33; где Dnmt3A представляет собой SEQ ID NO: 26 и Dnmt3L представляет собой SEQ ID NO: 28) в один полипептид. На Фиг. 1C представлены схематические изображения чувствительного к метилированию репортера GFP (адаптировано из Stelzer et al., Cell 2015), который был использован для оценки длительного сайленсинга, вызванного с помощью белка «все в одном». На Фиг. 1D-1E представлена диаграмма и результаты экспериментального рабочего процесса по технологии мутагенеза «hit-and-run» в клетках HEK293T, экспрессирующих репортер GFP, представленный на Фиг. 3. На Фиг. 1D показано, что в клетки были котрансфицированы плазмиды, одна из которых кодировала белок «hit-and-run», а другая плазмида кодировала егРНК. На Фиг. 1E показаны результаты анализа по технологии мутагенеза «hit-and-run», с сортировкой клеток, которые были котрансфицированы одновременно плазмидой «все в одном» и плазмидой егРНК. На Фиг. 1F показано, что результаты сайленсинга репортера GFP зависят от последовательности егРНК.
[0013] На Фиг. 2A-2F описан длительный сайленсинг эндогенных генов. На Фиг. 2A-2C представлены репрезентативные данные проточной цитометрии, полученные через 22 суток после трансфекции с последующим воздействием на гены (CD29, CD81, CD151), направленным на долгосрочный сайленсинг с использованием белка «все в одном». В квадранте IV представлены клетки, которые отключили соответствующий ген, на что указывает процент клеток с отключенным геном (т.е. 45%, 66% и 53%, соответственно). На Фиг. 2D представлена количественная оценка сайленсинга генов CD29, CD81 и CD151 с помощью трех различных егРНК. На Фиг. 2E представлена количественная оценка сайленсинга двух или трех генов одновременно, чтобы показать, что белок «все в одном» может быть мультиплексирован путем совместной доставки молекул егРНК, нацеленных на разные гены. На Фиг. 2F представлен график, который описывает момент времени, взятый через 9 месяцев после трансфекции белка «все в одном» и егРНК, нацеленной на ген CLTA, и который означает, что большинство клеток стабильно отключили ген CLTA.
[0014] На Фиг. 3A-3I описан долгосрочный сайленсинг эндогенных генов. На Фиг. 3A-3C показано, что собранные клетки утратили экспрессию CD29 (Фиг. 3A), CD81 (Фиг. 3B) и CD151 (Фиг. 3C) через тридцать шесть суток после трансфекции, как определено по их профилям экспрессии РНК. Фиг. 3D-3F представляют собой вулканные диаграммы, показывающие, что целевые гены CD29 (Фиг. 3D), CD81 (Фиг. 3E) и CD151 (Фиг. 3F) являются единственным значимым генным нокдауном для каждого эксперимента, что свидетельствует о высокой специфичности генного сайленсинга. На Фиг. 3G-3I представлена количественная оценка уровней транскриптов CD151 (Фиг. 3G), CD81 (Фиг. 3H) и CD29 (Фиг. 3I), демонстрирующая нокдаун более 96% каждого из целевых генов.
[0015] На Фиг. 4A-4H описан долгосрочный сайленсинг генов в различных линиях клеток млекопитающих. Фиг. 4A - 4F представляют собой графики проточной цитометрии, показывающие экспрессию синего флуоресцентного белка (СФБ, англ. «BFP») (который слит с белком «все в одном») в HeLa (цервикальные клетки) (Фиг. 4A), U2OS (клетки костной ткани) (Фиг. 4B) и в индуцированных плюрипотентных стволовых клетках человека (иПСК) (Фиг. 4С). Фиг. 4D-4F представляют собой нетрансфицированные контроли для Фиг. 4A-4C, соответственно. На Фиг. 4G показано, что был достигнут стабильный сайленсинг эндогенных генов в клетках HeLa и U2OS, измеренный через 18 суток после трансфекции белком «все в одном». На Фиг. 4A-4F по оси x отложен СФБ (слитый с белком «все в одном»), а по оси y - mCherry. На Фиг. 4H показано, что сайленсинг генов был обнаружен через 14 суток после трансфекции с помощью метода кПЦР в линиях клеток гепатоцитов AML12 мыши при нацеливании на Pcsk9, Npc1, Spcs1 и Cd81.
[0016] На Фиг. 5 представлены схемы слитых белков p76, p90 - p102 и p112, которые соответствуют последовательностям SEQ ID NO: 1-15, соответственно.
[0017] На Фиг. 6A-6E описаны действия по генному сайленсингу вариантов белка «все в одном». На Фиг. 6A-6B показаны результаты генного сайленсинга через 18 суток после трансфекции слитых белков последовательностей SEQ ID NO: 1-15, трансфицированных в клетки HEK293T для целенаправленного подавления гена CLTA. Форматы dCas9 - KRAB и dCas9 - Dnmt3A - Dnmt3L продемонстрировали кратковременную и более низкую эффективность длительного сайленсинга. На Фиг. 6C-6D представлено сравнение последовательностей SEQ ID NO: 1 (p76) и SEQ ID NO: 15 (p112) в сайленсинге эндогенного гена HIST2H2BE (H2B) (Фиг. 6C) и синтетического репортерного гена Snrpn-GFP (Фиг. 6D), стабильно экспрессируемых в клетках HEK293T. На Фиг. 6E представлен график экспрессии белков (пунктирные линии) p76 и p112 в течение периода времени в 50 суток для выключения гена HIST2H2BE (H2B). Уровни белка измеряли с помощью метода проточной цитометрии для детектирования СФБ, который экспрессируется совместно с белком «все в одном».
[0018] На Фиг. 7A-7B представлены вестерн-блоты вариантов белка «все в одном». На Фиг. 7А представлен анализ по методу вестерн-блоттинг вариантов белка «все в одном» p76 и p90 - p102 с использованием антитела против Steptococcus pyogenes Cas9. Верхняя полоса соответствует полноразмерному белку, а полосы меньшего размера отображают продукты протеолиза белков «все в одном». На Фиг. 7В представлен анализ по методу вестерн-блоттинг вариантов белка «все в одном» с целью обнаружения свободного Dnmt3A, который отщепляется от слитого белка.
[0019] На Фиг. 8A-8E описан объединенный скрининг для определения оптимальных егРНК. На Фиг. 8A представлена схема объединенного скрининга для определения оптимальных егРНК, которые приводят к долгосрочному сайленсингу генов. На Фиг. 8B-8E представлены гистограммы проточной цитометрии, показывающие процент клеток, подвергающихся сайленсингу генов через четыре недели после трансфекции. Были использованы четыре клеточные линии HEK293T, каждая с различным геном с меткой GFP, включительно с CLTA (Фиг. 8B), VIM (Фиг. 8C), HIST2H2BE (H2B) (Фиг. 8D) и RAB11A (Фиг. 8E).
[0020] На Фиг. 9A-9D представлены карты функциональности егРНК в сайте начала транскрипции целевого гена, включительно с CLTA (Фиг. 9A), H2B (Фиг. 9B), RAB11 (Фиг. 9C) и VIM (Фиг. 9D). Сайт начала транскрипции (СНТ, англ. «TSS») и островок CpG аннотированы над каждым графиком. Каждая точка отображает собой одну егРНК, а ее эффективность в долгосрочном генном сайленсинге отображено как изменение в log2-кратном содержании егРНК. Степень заполнения нуклеосом (нижний график) нанесена на графики по сигналу от МНазы (микрококковая нуклеаза, англ. «MNase»).
[0021] На Фиг. 10A-10E описаны функциональные егРНК для длительного сайленсинга генов. На Фиг. 10А представлен рабочий процесс объединенного скрининга в клетках HEK293T для определения оптимальных положений нацеливания егРНК для белка «все в одном», адаптированный из предыдущего рицинового скрининга на биочипах высокой плотности в клетках K562 для определения оптимальных егРНК для dCas9 - KRAB (Gilbert, Horlbeck et al., Cell 2014). На Фиг. 10B-10E представлены репрезентативные графики, показывающие фенотипы роста для четырех генов, включительно с ARL1 (Фиг. 10B), EIF6 (Фиг. 10C), SMC3 (Фиг. 10D), HEATR1 (Фиг. 10E), из существующих наборов данных для dCas9 - KRAB/ CRISPRi в клетках K562 (Gilbert, Horlbeck et al., 2014), и с белком «все в одном» (нижний график). Каждая точка отображает собой егРНК. СНТ и аннотированный островок CpG показаны для каждого гена.
[0022] На Фиг. 11A-11B представлено сравнение фенотипов роста и позиционирования нуклеосом (по сигналу от МНазы) для VPS53 (Фиг. 11A) и VPS54 (Фиг. 11B), которое показывает расположение функциональных егРНК в областях, лишенных нуклеосом.
[0023] На Фиг. 12A-12C показана доставка белка «все в одном» посредством экспрессии мРНК. На Фиг. 12A показана транскрипция in vitro двух вариантов белка «все в одном» (p102 и p112), а также показан полноразмерный синтез каждой конструкции. На Фиг. 12B представлен график проточной цитометрии, показывающий экспрессию p102 и p112 через одни сутки после трансфекции мРНК в клетки HEK293T. На Фиг. 12С представлена динамика сайленсинга эндогенного гена CLTA в клетках HEK293T после трансфекции мРНК, экспрессирующей варианты p102 и p112 белка «все в одном».
[0024] На Фиг. 13A-13G описана контролируемая экспрессия белка «все в одном» с помощью доксициклиновой индукции. На Фиг. 13A представлены графики проточной цитометрии, показывающие индуцированную экспрессию белка «все в одном» путем добавления доксициклина в клетки K562, которые стабильно кодируют белок «все в одном» под действием промотора, индуцируемого доксициклином. Пунктирная линия отображает базовую медианную флуоресценцию СФБ без введения доксициклина. На Фиг. 13B представлены вестерн-блоты клеток, направленные на детекцию экспрессии белка «все в одном» до и после обработки доксициклином. На Фиг. 13C-13F представлены графики проточной цитометрии для нокдауна генов CD81 (Фиг. 13C- 13D) и CD151 (Фиг. 13E-13F) через 14 суток после обработки доксициклином клеток K562. На Фиг. 13G представлена количественная оценка нокдауна генов CD81 и CD151 через 14 суток после обработки доксициклином или без обработки доксициклином.
ПОДРОБНОЕ ОПИСАНИЕ СУЩНОСТИ ИЗОБРЕТЕНИЯ
[0025] Описанная в данном документе технология позволяет, среди прочего, обеспечить постоянный сайленсинг генов в клетках млекопитающих без образования двухцепочечных разрывов ДНК в геноме хозяина. В вариантах реализации данного изобретения центральный компонент представляет собой одиночную полипептидную цепь, состоящую из каталитически неактивного Cas9 (dCas9), слитого с Dnmt3A, Dnmt3L, и доменом KRAB (в данном документе указанный центральный компонент именуется как «белок «все в одном»»). Указанный слитый белок, представленный в данном документе, может быть направлен на конкретный сайт в геноме млекопитающего с использованием единой гидовой РНК (егРНК), и может приводить к метилированию ДНК и (или) вносить репрессивные хроматиновые маркеры в этот сайт. В вариантах реализации данного изобретения результатом является сайленсинг гена, который наследуется при последующих делениях клеток. В вариантах реализации данного изобретения слитый белок, представленный в данном документе (и егРНК), экспрессируются только кратковременно, минуя использование способов вирусной доставки для индукции постоянного сайленсинга.
[0026] В вариантах реализации данного изобретения слитые белки, представленные в данном документе, обеспечивают надежный долгосрочный или постоянный сайленсинг экспрессии эндогенных генов посредством редактирования эпигенома, а не генома. Возможно как нацеливание на оба аллеля гена, так и селективное нацеливание на один патогенный аллель. В вариантах реализации данного изобретения преимущество слитого белка, представленного в данном документе, состоит в том, что эпигенетическое редактирование является обратимым и, следовательно, по своей природе более безопасным, чем редактирование генома. Таким образом, в вариантах реализации данного изобретения слитый белок, представленный в данном документе, полезен в профилактических целях. Например, сайленсинг гена может обеспечить острую защиту от инфекции/ биологического токсина, а затем сайленсинг может быть обращен вспять после отсутствия риска инфекции или интоксикации. Таким образом, в вариантах реализации данного изобретения слитый белок, представленный в данном документе, обладает полезными свойствами в ситуации, при которой вирус или токсин проникает в клетку посредством взаимодействия с белком, который требуется для долговременной функции органа или для гомеостаза. В вариантах реализации данного изобретения слитый белок, представленный в данном документе, полезен в терапевтических средствах, основанных на редактировании генома.
[0027] В вариантах реализации данного изобретения постоянный сайленсинг гена в клетках млекопитающих может быть достигнут с помощью двух компонентов: одной полипептидной цепи, состоящей из dCas9, слитого с тремя эпигенетическими модуляторами, и единой гидовой РНК, которая направляет белок в конкретный сайт в геноме хозяина. В вариантах реализации данного изобретения компоненты экспрессируются в клетке-хозяине только кратковременно, что снижает токсичность и нецелевые явления.
[0028] В вариантах реализации данного изобретения слитый белок, представленный в данном документе, не вызывает разрывов ДНК в клетке-хозяине для обеспечения постоянного генного сайленсинга. В вариантах реализации данного изобретения эпигенетические метки, которые добавляются к представляющему интерес геномному сайту, являются обратимыми, что позволяет удалять любые нецелевые явления, которые могут произойти.
[0029] Определения
[0030] Несмотря на то, что в данном документе показаны и описаны различные варианты реализации и аспекты данного изобретения, для специалистов в данной области техники будет очевидно, что такие варианты реализации и аспекты представлены только в качестве примера. Многочисленные вариации, изменения и замены теперь будут очевидны специалистам в данной области техники без отклонения от данного изобретения. Следует понимать, что при практическом применении изобретения могут использоваться различные альтернативы описанным в данном документе вариантам реализации данного изобретения.
[0031] Заголовки разделов, используемые в данном документе, предназначены только для организационных целей и не должны толковаться как ограничивающие описанный объект данного изобретения. Все документы или части документов, процитированные в данной заявке, включая, помимо прочего, патенты, заявки на патенты, статьи, книги, руководства и научные трактаты, настоящим полностью включены в данное описание посредством ссылки в полном объеме для любых целей.
[0032] Если не указано иное, все технические и научные термины, употребляемые в данном документе, имеют значение, обычно понятное специалисту в области техники, к которой принадлежит данное изобретение. Следующие ссылки предоставляют специалисту общее определение многих терминов, употребляемых в данном изобретении: Singleton et al., Dictionary of Microbiology and Molecular Biology (2nd ed. 1994); The Cambridge Dictionary of Science and Technology (Walker ed., 1988); The Glossary of Genetics, 5th Ed., R. Rieger et al. (eds.), Springer Verlag (1991); и Hale & Marham, The Harper Collins Dictionary of Biology (1991). В контексте данного документа нижеследующие термины имеют приписываемые им значения, если не указано иное.
[0033] Использование единственного или множественного числа в данном описании и в последующей формуле изобретения следует традиционному подходу в патентах, означающему «по меньшей мере один», за исключением ситуации, когда в конкретном случае из контекста ясно, что термин предназначен в этом конкретном случае для обозначения конкретно одного и только одного. Аналогичным образом, термин «содержащий» является неограничивающим, и не исключает дополнительных элементов, функций, компонентов и т.д. Литературные источники, указанные в данном документе, прямо включены в данный документ посредством ссылки во всем их объеме, если не указано иное.
[0034] Термины «содержать», «включать» и «иметь», а также их производные употребляются в контексте данного документа взаимозаменяемо как широкие неограничивающие термины. Например, при употреблении терминов «содержащий», «включающий» или «имеющий» подразумевается, что какой бы элемент ни содержался, был включен или имелся, это не единственный элемент, охватываемый подлежащим предложения, содержащим соответствующий указанный выше глагол.
[0035] Термин «нуклеиновая кислота» относится к нуклеотидам (например, дезоксирибонуклеотидам или рибонуклеотидам) и их полимерам в одноцепочечной, двухцепочечной или многоцепочечной форме, или к их комплементам. Термины «полинуклеотид», «олигонуклеотид», «олиго» и тому подобное относятся в обычном и общепринятом смысле к линейной последовательности нуклеотидов. Термин «нуклеотид» относится в обычном и общепринятом смысле к отдельной единице полинуклеотида, то есть к мономеру. Нуклеотиды могут быть рибонуклеотидами, дезоксирибонуклеотидами или их модифицированными версиями. Примеры полинуклеотидов, предусмотренных в данном документе, включают одно- и двухцепочечную ДНК, одно- и двухцепочечную РНК, и гибридные молекулы, содержащие смеси одно- и двухцепочечных ДНК и РНК. Примеры нуклеиновых кислот, например полинуклеотидов, предусмотренных в данном документе, включают, но не ограничиваются ими, любой тип РНК, например мРНК, миРНК, микроРНК, егРНК и гидовую РНК, а также любой тип ДНК, например геномную ДНК, плазмидную ДНК и миникольцевую ДНК, и любые их фрагменты. В аспектах данного изобретения нуклеиновая кислота представляет собой информационную РНК. В некоторых аспектах данного изобретения информационная РНК представляет собой информационный рибонуклеопротеин (РНП). Термин «дуплекс», используемый в данном документе в контексте полинуклеотидов, относится, в обычном и общепринятом смысле, к двухцепочечности. Нуклеиновые кислоты могут быть линейными или разветвленными. Например, нуклеиновые кислоты могут представлять собой линейную цепь нуклеотидов или нуклеиновые кислоты могут быть разветвленными, например, так, что нуклеиновые кислоты содержат одно или несколько плеч или разветвлений нуклеотидов. Необязательно, разветвленные нуклеиновые кислоты многократно разветвлены с образованием структур более высокого порядка, таких как дендримеры и тому подобное.
[0036] В контексте данного документа термины «нуклеиновая кислота», «молекула нуклеиновой кислоты», «олигомер нуклеиновой кислоты», «олигонуклеотид», «последовательность нуклеиновой кислоты», «фрагмент нуклеиновой кислоты» и «полинуклеотид» употребляются взаимозаменяемо и предназначены для включения, но не ограничиваясь ими, полимерной формы нуклеотидов, ковалентно связанных вместе, которые могут иметь различную длину, либо дезоксирибонуклеотидов, либо рибонуклеотидов, или их аналогов, производных или модификаций. Различные полинуклеотиды могут иметь разные трехмерные структуры и могут выполнять разнообразные функции, известные или неизвестные. Неограничивающие примеры полинуклеотидов включают в себя ген, фрагмент гена, экзон, интрон, межгенную ДНК (включая, без ограничения, гетерохроматическую ДНК), информационную РНК (иРНК), транспортную РНК, рибосомную РНК, рибозим, кДНК, рекомбинантный полинуклеотид, разветвленный полинуклеотид, плазмиду, вектор, выделенную ДНК последовательности, выделенную РНК последовательности, егРНК, гидовую РНК, зонд нуклеиновой кислоты и праймер. Полинуклеотиды, используемые в способах согласно данному изобретению, могут содержать природные последовательности нуклеиновых кислот и их варианты, искусственные последовательности нуклеиновых кислот, или комбинацию таких последовательностей.
[0037] Полинуклеотид обычно состоит из определенной последовательности четырех нуклеотидных оснований: аденина (А); цитозина (С); гуанина (G); и тимина (T) (урацила (U) вместо тимина (T), когда полинуклеотид представляет собой РНК). Таким образом, термин «полинуклеотидная последовательность» представляет собой алфавитное представление полинуклеотидной молекулы; альтернативно, этот термин может применяться к самой молекуле полинуклеотида. Такое алфавитное представление может вводиться в базы данных на компьютере, имеющем центральный процессор, и иметь биоинформационное применение, такое как использование в функциональной геномике и поиск гомологий. Полинуклеотиды могут необязательно включать в себя один или несколько нестандартных нуклеотидов, нуклеотидных аналогов и (или) модифицированных нуклеотидов.
[0038] Нуклеиновые кислоты, включая, например, нуклеиновые кислоты с фосфотиоатным остовом, могут включать в себя один или несколько реактивных фрагментов. Употребляемый в контексте данного документа термин «реактивный фрагмент» охватывает собой любую группу, способную реагировать с другой молекулой, например, нуклеиновой кислотой или полипептидом, посредством ковалентных, нековалентных или других взаимодействий. В качестве примера нуклеиновая кислота может включать в себя фрагмент, реакционноспособный по отношению к аминокислоте, который вступает в реакцию с аминокислотой на белке или полипептиде посредством ковалентного, нековалентного или другого взаимодействия.
[0039] Термины также охватывают нуклеиновые кислоты, содержащие известные аналоги нуклеотидов или модифицированные остатки или связи основной цепи, которые являются синтетическими, встречающимися в природе и не встречающимися в природе, которые имеют такие же связывающие свойства, что и референсная нуклеиновая кислота, и которые метаболизируются аналогично референсным нуклеотидам. Примеры таких аналогов включают, но не ограничиваются ими, производные фосфодиэфиров, включая, например, фосфорамидат, фосфородиамидат, фосфоротиоат (также известный как фосфотиоат, содержащий посредством двойной связи серу, которая заместила кислород в фосфате), фосфородитиоат, фосфонокарбоновые кислоты, фосфонокарбоксилаты, фосфоноуксусные кислоты, фосфоноформовую кислоту, метилфосфонатные, борфосфонатные или O-метилфосфорамидитные связи (см. Eckstein, Oligonucleotides and Analogues: A Practical Approach, Oxford University Press), а также модификации нуклеотидных оснований, такие как 5-метилцитидин или псевдоуридин; и остовы и связи пептидных нуклеиновых кислот. Другие аналоги нуклеиновых кислот включают в себя нуклеиновые кислоты с положительным остовом; неионные остовы, модифицированные сахара и не рибозные остовы (например, фосфородиамидатные морфолиновые олигонукледотиды или закрытые нуклеиновые кислоты (ЗНК, англ. «LNA»), как известно в данной области техники), включая те, что описаны в патентах США № 5235033 и № 5034506, а также в Главах 6 и 7 из ASC Symposium Series 580, Carbohydrate Modifications in Antisense Research, Sanghui & Cook, eds. Нуклеиновые кислоты, содержащие один или несколько карбоциклических сахаров, также включены в одно определение нуклеиновых кислот. Модификации рибозофосфатного остова могут осуществляться по разным причинам, например, для повышения стабильности и периода полужизни таких молекул в физиологической среде или в качестве зондов на биочипе. Могут быть приготовлены смеси встречающихся в природе нуклеиновых кислот и аналогов; альтернативно могут быть приготовлены смеси различных аналогов нуклеиновых кислот и смеси встречающихся в природе нуклеиновых кислот и аналогов. В аспектах данного изобретения межнуклеотидные связи в ДНК представляют собой фосфодиэфирные связи, производные фосфодиэфирных связей, или их комбинацию.
[0040] Нуклеиновые кислоты могут включать в себя неспецифические последовательности. Употребляемый в контексте данного документа термин «неспецифическая последовательность» относится к последовательности нуклеиновой кислоты, которая содержит серию остатков, не предназначенных для комплементарности любой другой последовательности нуклеиновой кислоты, или только частично комплементарных ей. В качестве примера, неспецифическая последовательность нуклеиновой кислоты представляет собой последовательность остатков нуклеиновой кислоты, которая не функционирует как ингибирующая нуклеиновая кислота при контакте с клеткой или организмом.
[0041] Термин «комплементарный» или «комплементарность» относится к способности нуклеиновой кислоты образовывать с другой последовательностью нуклеиновой кислоты водородную связь (водородные связи) либо традиционного типа по Уотсону-Крику, либо других нетрадиционных типов. Например, последовательность A - G - T комплементарна последовательности T - C - A. Процент комплементарности указывает процент остатков в молекуле нуклеиновой кислоты, которые могут образовывать водородные связи (например, спаривание оснований по Уотсону-Крику) со второй последовательностью нуклеиновой кислоты (например, 5, 6, 7, 8, 9, 10 из 10 являются комплементарными на 50%, 60%, 70%, 80%, 90% и 100%, соответственно). Термин «совершенно комплементарный» означает, что все смежные остатки последовательности нуклеиновой кислоты будут связываться водородными связями с тем же числом смежных остатков во второй последовательности нуклеиновой кислоты. Употребляемый в контексте данного документа термин «по существу комплементарный» относится к степени комплементарности, которая составляет по меньшей мере 60%, 65%, 70%, 75%, 80%, 85%, 90%, 95%, 97%, 98%, 99% или 100% в участке из 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 30, 35, 40, 45, 50 или более нуклеотидов, или относится к двум нуклеиновым кислотам, которые гибридизуются в жестких условиях (т.е. в жестких условиях гибридизации).
[0042] Фраза «жесткие условия гибридизации» относится к условиям, при которых зонд будет гибридизоваться со своей целевой подпоследовательностью, обычно в сложной смеси нуклеиновых кислот, но не с другими последовательностями. Жесткие условия зависят от последовательности и будут разными в разных обстоятельствах. Более длинные последовательности специфически гибридизуются при более высоких температурах. Подробное руководство по гибридизации нуклеиновых кислот можно найти в Tijssen, Techniques in Biochemistry and Molecular Biology - Hybridization with Nucleic Probes, “Overview of principles of hybridization and the strategy of nucleic acid assays” (1993). Обычно выбираются жесткие условия, которые примерно на 5-10°C ниже, чем температура точки плавления (Tпл) для конкретной последовательности при определенной ионной силе и pH. Tпл представляет собой такую температуру (при определенной ионной силе, pH и концентрации нуклеиновых кислот), при которой 50% зондов, комплементарных мишени, гибридизуются с последовательностью-мишенью при равновесном состоянии (так как последовательности-мишени присутствуют в избытке, то при Tпл 50% зондов заняты при равновесном состоянии). Жесткие условия также могут быть достигнуты путем добавления дестабилизирующих агентов, таких как формамид. Для селективной или специфической гибридизации положительный сигнал должен по крайней мере в два раза превышать фон, предпочтительно - в 10 раз выше фоновой гибридизации. Примеры жестких условий гибридизации могут быть следующими: 50% формамид, 5x ССР (стандартный солевой раствор, англ. «SSC») и 1% ДСН, инкубирование при температуре 42°C, или 5x ССР, 1% ДСН, инкубирование при температуре 65°C, с промывкой в 0,2x ССР и 0,1% ДСН при температуре 65°C.
[0043] Нуклеиновые кислоты, которые не гибридизуются друг с другом в жестких условиях, все же по существу идентичны, если полипептиды, которые кодируются ими, по существу идентичны. Это происходит, например, когда создается копия нуклеиновой кислоты с использованием максимальной вырожденности кодонов, разрешенной генетическим кодом. В таких случаях нуклеиновые кислоты обычно гибридизуются в умеренно жестких условиях гибридизации. Примеры «умеренно жестких условий гибридизации» включают в себя гибридизацию в буфере, содержащем 40% формамида, 1 М NaCl, 1% ДСН при температуре 37°C и промывку в 1X ССР при температуре 45°C. Положительная гибридизация представляет собой такую, которая по меньшей мере вдвое превышает фоновую гибридизацию. Рядовой специалист в данной области техники легко поймет, что можно использовать альтернативные условия гибридизации и промывки для обеспечения условий похожей жесткости. Дополнительные рекомендации по определению параметров гибридизации представлены в многочисленных ссылках, например, Current Protocols in Molecular Biology, ed. Ausubel, et al., см. выше.
[0044] Термин «ген» означает сегмент ДНК, участвующий в производстве белка; он включает в себя области, предшествующие и следующие за кодирующей областью (лидирующую и отстающую), а также промежуточные последовательности (интроны) между отдельными кодирующими сегментами (экзонами). Лидирующая и отстающая области, так же как и интроны, включают в себя регуляторные элементы, которые необходимы во время транскрипции и трансляции гена. Кроме того, «белковый генный продукт» представляет собой белок, экспрессируемый конкретным геном.
[0045] Слово «экспрессия» или «экспрессируемый», используемое в контексте данного документа по отношению к гену, означает продукт транскрипции и (или) трансляции этого гена. Уровень экспрессии молекулы ДНК в клетке может быть определен либо на основании количества соответствующей иРНК, которая присутствует в клетке, либо на основании количества белка, кодируемого этой ДНК, продуцируемого клеткой. Уровень экспрессии некодирующих молекул нуклеиновых кислот (например, егРНК) может быть определен стандартными методами ПЦР или нозерн-блоттингом, хорошо известными в данной области техники. См. Sambrook et al., 1989 Molecular Cloning: A Laboratory Manual, 18.1-18.88.
[0046] Термин «регуляторная последовательность транскрипции», употребляемый в контексте данного документа, относится к сегменту ДНК, который способен повышать или понижать транскрипцию (например, экспрессию) конкретного гена в организме. Неограничивающие примеры регуляторных последовательностей транскрипции включают в себя промоторы, энхансеры и сайленсеры.
[0047] Термины «сайт начала транскрипции» и «сайт инициации транскрипции» могут употребляться взаимозаменяемо для обозначения в контексте данного документа 5'-конца последовательности гена (например, последовательности ДНК), где РНК-полимераза (например, ДНК-направленная РНК-полимераза) начинает синтез транскрипта РНК. Сайт начала транскрипции может быть первым нуклеотидом транскрибируемой последовательности ДНК, где РНК-полимераза начинает синтезировать транскрипт РНК. Квалифицированный специалист в данной области техники может определить сайт начала транскрипции с помощью рутинных экспериментов и анализа, например, путем выполнения анализа по «прерванной транскрипции», или с помощью определений в соответствии с базой данных FANTOM5.
[0048] Термин «промотор», употребляемый в контексте данного документа, относится к области ДНК, которая инициирует транскрипцию конкретного гена. Промоторы обычно расположены рядом с сайтом начала транскрипции гена, выше гена и на одной и той же цепи ДНК (т.е. в положении 5' на смысловой цепи ДНК). Промоторы могут иметь длину от около 100 до около 1000 пар оснований.
[0049] Термин «энхансер», употребляемый в контексте данного документа, относится к области ДНК, которая может быть связана белками (например, факторами транскрипции) для увеличения вероятности того, что произойдет транскрипция гена. Энхансеры могут иметь длину от около 50 до около 1500 пар оснований. Энхансеры могут быть расположены ниже или выше сайта инициации транскрипции, который они регулируют, и могут находиться на расстоянии нескольких сотен пар оснований от сайта инициации транскрипции.
[0050] Термин «сайленсер», употребляемый в контексте данного документа, относится к последовательности ДНК, которая способна связывать факторы регуляции транскрипции, известные как репрессоры, тем самым отрицательно влияя на транскрипцию гена. Последовательности сайленсеров ДНК могут быть обнаружены во многих различных положениях по всей длине ДНК, включая, но не ограничиваясь этим, перед целевым геном, в отношении которого они действуют путем подавления транскрипции гена (например, вызывая сайленсинг экспрессии гена).
[0051] Термин «гидовая РНК» или «гРНК», употребляемый в контексте данного документа, относится к любой полинуклеотидной последовательности, имеющей достаточную комплементарность с целевой полинуклеотидной последовательностью для гибридизации с целевой последовательностью и прямого последовательность-специфичного связывания комплекса CRISPR с целевой последовательностью. В аспектах данного изобретения степень комплементарности между гидовой последовательностью и соответствующей ей целевой последовательностью при оптимальном выравнивании с использованием подходящего алгоритма выравнивания составляет около или более чем около 50%, 60%, 75%, 80%, 85%, 90%, 95%, 97,5%, 99%, или более.
[0052] В вариантах реализации данного изобретения полинуклеотид (например, гРНК) представляет собой одноцепочечную рибонуклеиновую кислоту. В аспектах данного изобретения полинуклеотид (например, гРНК) имеет длину 10, 20, 30, 40, 50, 60, 70, 80, 90, 100 или более остатков нуклеиновой кислоты. В аспектах данного изобретения полинуклеотид (например, гРНК) имеет длину от 10 до 30 остатков нуклеиновой кислоты. В аспектах данного изобретения полинуклеотид (например, гРНК) имеет длину 20 остатков нуклеиновой кислоты. В аспектах данного изобретения длина полинуклеотида (например, гРНК) может составлять по меньшей мере 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39, 40, 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51, 52, 53, 54, 55, 56, 57, 58, 59, 60, 61, 62, 63, 64, 65, 66, 67, 68, 69, 70, 71, 72, 73, 74, 75, 76, 77, 78, 79, 80, 81, 82, 83, 84, 85, 86, 87, 88, 89, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99, 100 или более остатков нуклеиновой кислоты или остатков сахара в длину. В аспектах данного изобретения полинуклеотид (например, гРНК) имеет длину от 5 до 50, от 10 до 50, от 15 до 50, от 20 до 50, от 25 до 50, от 30 до 50, от 35 до 50, от 40 до 50, от 45 до 50, от 5 до 75, от 10 до 75, от 15 до 75, 20 до 75, от 25 до 75, от 30 до 75, от 35 до 75, от 40 до 75, от 45 до 75, от 50 до 75, от 55 до 75, от 60 до 75, от 65 до 75, от 70 до 75, от 5 до 100, от 10 до 100, От 15 до 100, от 20 до 100, от 25 до 100, от 30 до 100, от 35 до 100, от 40 до 100, от 45 до 100, от 50 до 100, от 55 до 100, от 60 до 100, от 65 до 100, от 70 до 100, 75 до 100, от 80 до 100, от 85 до 100, от 90 до 100, от 95 до 100 или более остатков. В аспектах данного изобретения полинуклеотид (например, гРНК) имеет длину от 10 до 15, от 10 до 20, от 10 до 30, от 10 до 40 или от 10 до 50 остатков.
[0053] Термин «аминокислота» относится к встречающимся в природе и синтетическим аминокислотам, а также к аналогам аминокислот и миметикам аминокислот, которые действуют аналогично встречающимся в природе аминокислотам. Встречающиеся в природе аминокислоты представляют собой такие аминокислоты, которые кодируются генетическим кодом, а также те аминокислоты, которые позже модифицируются, например, гидроксипролин, γ-карбоксиглутамат и О-фосфосерин. Аналоги аминокислот относятся к соединениям, которые имеют ту же базовую химическую структуру, что и встречающиеся в природе аминокислоты, то есть α-углерод, связанный с водородом, карбоксильную группу, аминогруппу и группу R, например, гомосерин, норлейцин, сульфоксид метионина, метилсульфоний метионина. Такие аналоги имеют модифицированные группы R (например, норлейцин) или модифицированные пептидные остовы, но сохраняют ту же базовую химическую структуру, что и встречающаяся в природе аминокислота. Миметики аминокислот относятся к химическим соединениям, которые имеют структуру, отличную от общей химической структуры аминокислоты, но которые действуют путем, похожим на таковой для встречающихся в природе аминокислот. Термины «не встречающаяся в природе аминокислота» и «неприродная аминокислота» относятся к аналогам аминокислот, синтетическим аминокислотам и миметикам аминокислот, которые не встречаются в природе.
[0054] Аминокислоты могут обозначаться в данном документе либо их общеизвестными трехбуквенными символами, либо однобуквенными символами, рекомендованными Комиссией по биохимической номенклатуре IUPAC-IUB. Нуклеотиды также могут обозначаться их общепринятыми однобуквенными кодами.
[0055] Термины «полипептид», «пептид» и «белок» употребляются в контексте данного документа взаимозаменяемо для обозначения полимера из аминокислотных остатков, при этом полимер, в аспектах данного изобретения, может быть конъюгирован с фрагментом, который не состоит из аминокислот. Данные термины применяются к аминокислотным полимерам, в которых один или несколько аминокислотных остатков являются искусственным химическим миметиком соответствующей встречающейся в природе аминокислоты, а также к встречающимся в природе аминокислотным полимерам и не встречающимся в природе аминокислотным полимерам. Термин «слитый белок» относится к химерному белку, кодирующему две или более отдельных белковых последовательностей, которые рекомбинантно экспрессируются как единый фрагмент.
[0056] Термин «консервативно модифицированные варианты» применяется к последовательностям как аминокислот, так и нуклеиновых кислот. Что касается конкретных последовательностей нуклеиновых кислот, «консервативно модифицированные варианты» относятся к тем нуклеиновым кислотам, которые кодируют идентичные или практически идентичные аминокислотные последовательности. Из-за вырожденности генетического кода любой данный белок будет кодироваться рядом последовательностей нуклеиновых кислот. Например, все кодоны GCA, GCC, GCG и GCU кодируют аминокислоту аланин. Таким образом, в каждом положении, в котором аланин определяется кодоном, этот кодон может быть изменен на любой из соответствующих описанных кодонов без изменения кодируемого полипептида. Такие вариации нуклеиновой кислоты представляют собой «молчащие вариации», которые представляют собой один из видов консервативно модифицированных вариаций. Каждая последовательность нуклеиновой кислоты в данном документе, которая кодирует полипептид, также описывает все возможные молчащие вариации нуклеиновой кислоты. Специалист поймет, что каждый кодон в нуклеиновой кислоте (за исключением AUG, который обычно является единственным кодоном для метионина, и TGG, который обычно является единственным кодоном для триптофана) может быть модифицирован с образованием функционально идентичной молекулы. Соответственно, присутствие каждой молчащей вариации нуклеиновой кислоты, кодирующей полипептид, подразумевается в каждой описанной последовательности.
[0057] Что касается аминокислотных последовательностей, специалист в данной области техники поймет, что отдельные замены, делеции или добавления к последовательности нуклеиновой кислоты, пептида, полипептида или белка, которые изменяют, добавляют или удаляют одну аминокислоту или небольшой процент аминокислот в кодируемой последовательность, создают тем самым «консервативно модифицированный вариант», в котором изменение приводит к замене аминокислоты на химически подобную аминокислоту. Таблицы консервативных замен, содержащие функционально похожие аминокислоты, хорошо известны в данной области техники. Такие консервативно модифицированные варианты дополняют и не исключают полиморфные варианты, межвидовые гомологи и аллели из данного описания. Каждая из следующих восьми групп содержит аминокислоты, которые являются консервативными заменами друг друга: (1) аланин (A), глицин (G); (2) аспарагиновая кислота (D), глутаминовая кислота (E); (3) аспарагин (N), глутамин (Q); (4) аргинин (R), лизин (K); (5) изолейцин (I), лейцин (L), метионин (M), валин (V); (6) фенилаланин (F), тирозин (Y), триптофан (W); (7) серин (S), треонин (T); и (8) цистеин (C), метионин (M) (см., например, Creighton, Proteins (1984)).
[0058] «Процент идентичности последовательностей» определяется путем сравнения двух оптимально выровненных последовательностей в окне сравнения, при этом часть полинуклеотидной или полипептидной последовательности в окне сравнения может включать в себя добавления или делеции (т.е. гэпы) по сравнению с референсной последовательностью (которая не содержит добавлений или делеций) для оптимального выравнивания двух последовательностей. Указанный процент рассчитывается путем определения количества положений, в которых идентичное основание нуклеиновой кислоты или аминокислотный остаток встречается в обеих последовательностях, чтобы получить количество совпадающих положений, путем деления количества совпадающих положений на общее количество положений в окне сравнения и умножением результата на 100, чтобы получить процент идентичности последовательностей.
[0059] Термины «идентичный» или процент «идентичности» в контексте двух или более нуклеиновых кислот или полипептидных последовательностей относятся к двум или более последовательностям или подпоследовательностям, которые являются одинаковыми или имеют определенный процент аминокислотных остатков или нуклеотидов, которые являются одинаковыми (т.е. идентичность составляет около 60%, предпочтительно 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более высокая идентичность в указанной области при сравнении и выравнивании для максимального соответствия в окне сравнения или в назначенной области) при измерении с использованием алгоритмов сравнения последовательностей BLAST или BLAST 2.0 с параметрами по умолчанию, которые описаны ниже, или путем ручного выравнивания и визуального осмотра (см., например, веб-сайт NCBI ncbi.nlm.nih.gov/BLAST/ или ему подобные). Такие последовательности далее именуются «по существу идентичными». Данное определение также относится или может применяться к комплементу тестовой последовательности. Данное определение также включает последовательности с делециями и (или) добавлениями, а также последовательности с заменами. Как описано ниже, предпочтительные алгоритмы могут учитывать гэпы и тому подобное. Предпочтительно, идентичность существует в области, длина которой составляет, по меньшей мере, около 25 аминокислот или нуклеотидов, или, более предпочтительно, в области, длина которой составляет 50-100 аминокислот или нуклеотидов.
[0060] «Положение» аминокислоты или нуклеотидного основания обозначается числом, которое последовательно идентифицирует каждую аминокислоту (или нуклеотидное основание) в референсной последовательности на основе ее положения относительно N-конца (или 5'-конца). Из-за делеций, вставок, усечений, слияний и тому подобного, которые необходимо учитывать при определении оптимального выравнивания, в целом номер аминокислотного остатка в тестовой последовательности, определяемый простым подсчетом от N-конца, не обязательно будет тем же, что и номер соответствующего положения в референсной последовательности. Например, в случае, когда вариант имеет делецию относительно выровненной референсной последовательности, в этом варианте в сайте делеции не будет аминокислоты, которая соответствует положению в референсной последовательности. Если имеется вставка в выровненной референсной последовательности, эта вставка не будет соответствовать нумерованному положению аминокислоты в референсной последовательности. В случае усечения или слияния могут быть отрезки аминокислот либо в референсной, либо в выровненной последовательности, которые не соответствуют какой-либо аминокислоте в соответствующей последовательности.
[0061] Термины «пронумерованная со ссылкой на» или «соответствующая» при использовании в контексте нумерации конкретной аминокислотной или полинуклеотидной последовательности относятся к нумерации остатков указанной референсной последовательности, когда данная аминокислотная или полинуклеотидная последовательность сравнивается с референсной последовательностью.
[0062] Для конкретных белков, описанных в данном документе (например, KRAB, dCas9, Dnmt3A, Dnmt3L), названный белок включает в себя любую из встречающихся в природе форм белка, либо варианты или гомологи, которые сохраняют активность названного белка (например, в пределах по меньшей мере 50%, 80%, 90%, 95%, 96%, 97%, 98%, 99% или 100% активности по сравнению с нативным белком). В аспектах данного изобретения варианты или гомологи по меньшей мере на 50%, на 55%, на 60%, на 65%, на 70%, на 75%, на 80%, на 85%, на 90%, на 95%, на 96%, на 97%, на 98%, на 99% или на 100% идентичны аминокислотной последовательности по всей длине последовательности, или на участке последовательности (например, на непрерывном участке длиной 50, 100, 150 или 200 аминокислот) по сравнению с формой, встречающейся в природе. В аспектах данного изобретения белок представляет собой белок, идентифицированный с помощью ссылки на его последовательность в NCBI. В аспектах данного изобретения белок представляет собой белок, идентифицированный с помощью ссылки на его последовательность в NCBI, или по функциональному фрагменту такой последовательности, или по ее гомологу.
[0063] Термин «домен, связанный с боксом  » или «домен KRAB», употребляемый в контексте данного документа, относится к категории доменов, репрессирующих транскрипцию, которые присутствуют примерно в 400 человеческих факторах транскрипции, основанных на белке «цинковые пальцы». Домены KRAB обычно включают в себя от около 45 до около 75 аминокислотных остатков. Описание доменов KRAB, включительно с их функцией и применением, можно найти, например, в Ecco, G., Imbeault, M., Trono, D., KRAB zinc finger proteins, Development 144, 2017; Lambert et al. The human transcription factors, Cell 172, 2018; Gilbert et al., Cell (2013); и Gilbert et al., Cell (2014), все из которых включены в данный документ посредством ссылки в полном объеме. В аспектах данного изобретения домен KRAB представляет собой домен KRAB Kox 1. В аспектах данного изобретения домен KRAB включает в себя последовательность, изложенную в SEQ ID NO: 16. В аспектах данного изобретения домен KRAB представляет собой последовательность с SEQ ID NO: 16. В аспектах данного изобретения домен KRAB включает в себя аминокислотную последовательность, которая по меньшей мере на 50%, на 55%, на 60%, на 65%, на 70%, на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98%, на 99% или на 100% идентична последовательности с SEQ ID NO: 16. В аспектах данного изобретения домен KRAB включает в себя аминокислотную последовательность, которая по меньшей мере на 75% идентична последовательности с SEQ ID NO: 16. В аспектах данного изобретения домен KRAB включает в себя аминокислотную последовательность, которая по меньшей мере на 80% идентична последовательности с SEQ ID NO: 16. В аспектах данного изобретения домен KRAB включает в себя аминокислотную последовательность, которая по меньшей мере на 85% идентична последовательности с SEQ ID NO: 16. В аспектах данного изобретения домен KRAB включает в себя аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 16. В аспектах данного изобретения домен KRAB включает в себя аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 16.
» или «домен KRAB», употребляемый в контексте данного документа, относится к категории доменов, репрессирующих транскрипцию, которые присутствуют примерно в 400 человеческих факторах транскрипции, основанных на белке «цинковые пальцы». Домены KRAB обычно включают в себя от около 45 до около 75 аминокислотных остатков. Описание доменов KRAB, включительно с их функцией и применением, можно найти, например, в Ecco, G., Imbeault, M., Trono, D., KRAB zinc finger proteins, Development 144, 2017; Lambert et al. The human transcription factors, Cell 172, 2018; Gilbert et al., Cell (2013); и Gilbert et al., Cell (2014), все из которых включены в данный документ посредством ссылки в полном объеме. В аспектах данного изобретения домен KRAB представляет собой домен KRAB Kox 1. В аспектах данного изобретения домен KRAB включает в себя последовательность, изложенную в SEQ ID NO: 16. В аспектах данного изобретения домен KRAB представляет собой последовательность с SEQ ID NO: 16. В аспектах данного изобретения домен KRAB включает в себя аминокислотную последовательность, которая по меньшей мере на 50%, на 55%, на 60%, на 65%, на 70%, на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98%, на 99% или на 100% идентична последовательности с SEQ ID NO: 16. В аспектах данного изобретения домен KRAB включает в себя аминокислотную последовательность, которая по меньшей мере на 75% идентична последовательности с SEQ ID NO: 16. В аспектах данного изобретения домен KRAB включает в себя аминокислотную последовательность, которая по меньшей мере на 80% идентична последовательности с SEQ ID NO: 16. В аспектах данного изобретения домен KRAB включает в себя аминокислотную последовательность, которая по меньшей мере на 85% идентична последовательности с SEQ ID NO: 16. В аспектах данного изобретения домен KRAB включает в себя аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 16. В аспектах данного изобретения домен KRAB включает в себя аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 16.
[0064] Термин «метилтрансфераза ДНК», употребляемый в контексте данного документа, относится к ферменту, который катализирует перенос метильной группы на ДНК. Неограничивающие примеры метилтрансфераз ДНК включают в себя Dnmt1, Dnmt3A, Dnmt3B и Dnmt3L. В аспектах данного изобретения метилтрансфераза ДНК представляет собой бактериальную цитозин-метилтрансферазу и (или) бактериальную нецитозин-метилтрансферазу. В зависимости от конкретной метилтрансферазы ДНК метилируются разные участки ДНК. Например, Dnmt3A для метилирования обычно нацелен на динуклеотиды CpG. Посредством метилирования ДНК метилтрансферазы ДНК могут изменять активность сегмента ДНК (например, экспрессию гена) без изменения последовательности ДНК. В аспектах данного изобретения метилирование ДНК приводит к репрессии транскрипции гена и (или) модуляции факторов транскрипции, чувствительных к метилированию, или CTCF. Как описано в данном документе, слитые белки могут включать в себя одну или несколько (например, две) метилтрансфераз ДНК. Когда метилтрансфераза ДНК включена как часть слитого белка, метилтрансфераза ДНК может называться «доменом метилтрансферазы ДНК». В аспектах данного изобретения домен метилтрансферазы ДНК включает в себя одну или несколько метилтрансфераз ДНК. В аспектах данного изобретения домен метилтрансферазы ДНК включает в себя две метилтрансферазы ДНК. В аспектах данного изобретения домен метилтрансферазы ДНК представляет собой Dnmt3A. В аспектах данного изобретения домен метилтрансферазы ДНК имеет аминокислотную последовательность с SEQ ID NO: 26. В аспектах данного изобретения домен метилтрансферазы ДНК имеет аминокислотную последовательность, которая по меньшей мере на 50%, на 55%, на 60%, на 65%, на 70%, на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98%, на 99% или на 100% идентична последовательности с SEQ ID NO: 26. В аспектах данного изобретения домен метилтрансферазы ДНК имеет аминокислотную последовательность, которая по меньшей мере на 75% идентична последовательности с SEQ ID NO: 26. В аспектах данного изобретения домен метилтрансферазы ДНК имеет аминокислотную последовательность, которая по меньшей мере на 80% идентична последовательности с SEQ ID NO: 26. В аспектах данного изобретения домен метилтрансферазы ДНК имеет аминокислотную последовательность, которая по меньшей мере на 85% идентична последовательности с SEQ ID NO: 26. В аспектах данного изобретения домен метилтрансферазы ДНК имеет аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 26. В аспектах данного изобретения домен метилтрансферазы ДНК имеет аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 26. В аспектах данного изобретения домен метилтрансферазы ДНК представляет собой Dnmt3L. В аспектах данного изобретения домен метилтрансферазы ДНК имеет аминокислотную последовательность с SEQ ID NO: 28. В аспектах данного изобретения домен метилтрансферазы ДНК имеет аминокислотную последовательность, которая по меньшей мере на 50%, на 55%, на 60%, на 65%, на 70%, на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98%, на 99% или на 100% идентична последовательности с SEQ ID NO: 28. В аспектах данного изобретения домен метилтрансферазы ДНК имеет аминокислотную последовательность, которая по меньшей мере на 75% идентична последовательности с SEQ ID NO: 28. В аспектах данного изобретения домен метилтрансферазы ДНК имеет аминокислотную последовательность, которая по меньшей мере на 80% идентична последовательности с SEQ ID NO: 28. В аспектах данного изобретения домен метилтрансферазы ДНК имеет аминокислотную последовательность, которая по меньшей мере на 85% идентична последовательности с SEQ ID NO: 28. В аспектах данного изобретения домен метилтрансферазы ДНК имеет аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 28. В аспектах данного изобретения домен метилтрансферазы ДНК имеет аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 28. В аспектах данного изобретения домен метилтрансферазы ДНК включает в себя Dnmt3A и Dnmt3L. В аспектах данного изобретения домен метилтрансферазы ДНК имеет аминокислотную последовательность с SEQ ID NO: 33. В аспектах данного изобретения домен метилтрансферазы ДНК имеет аминокислотную последовательность, которая по меньшей мере на 50%, на 55%, на 60%, на 65%, на 70%, на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98%, на 99% или на 100% идентична последовательности с SEQ ID NO: 33. В аспектах данного изобретения домен метилтрансферазы ДНК имеет аминокислотную последовательность, которая по меньшей мере на 75% идентична последовательности с SEQ ID NO: 33. В аспектах данного изобретения домен метилтрансферазы ДНК имеет аминокислотную последовательность, которая по меньшей мере на 80% идентична последовательности с SEQ ID NO: 33. В аспектах данного изобретения домен метилтрансферазы ДНК имеет аминокислотную последовательность, которая по меньшей мере на 85% идентична последовательности с SEQ ID NO: 33. В аспектах данного изобретения домен метилтрансферазы ДНК имеет аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 33. В аспектах данного изобретения домен метилтрансферазы ДНК имеет аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 33. Описание структуры домена Dnmt3A-3L и его применения можно найти, например, в Siddique et al, Targeted methylation and gene silencing of VEGF-A in human cells by using a designed Dnmt3a-Dnmt3L single-chain fusion protein with increased DNA methylation activity, J. Mol. Biol. 425, 2013, и в Stepper et al, Efficient targeted DNA methylation with chimeric dCas9-Dnmt3a-Dnmt3L methyltransferase, Nucleic Acids Res. 45, 2017, которые полностью и для всех целей включены в данный документ посредством ссылки.
[0065] Белки «Dnmt3A», «Dnmt3a», «ДНК (цитозин-5)-метилтрансфераза 3A» или «метилтрансфераза ДНК 3a», указанные в данном документе, включают в себя любую из рекомбинантных или встречающихся в природе форм фермента Dnmt3A, либо его варианты или гомологи, которые сохраняют активность фермента Dnmt3A (например, в пределах по меньшей мере 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% активности по сравнению с Dnmt3A). В аспектах данного изобретения варианты или гомологи по меньшей мере на 90%, на 95%, на 96%, на 97%, на 98%, на 99% или на 100% идентичны аминокислотной последовательности по всей длине последовательности, или на участке последовательности (например, на непрерывном участке длиной 50, 100, 150 или 200 аминокислот) по сравнению с белком Dnmt3A, встречающимся в природе. В аспектах данного изобретения белок Dnmt3A по существу идентичен белку, идентифицированному по регистрационному номеру UniProt Q9Y6K1, либо варианту или гомологу, по существу идентичному белку с данным регистрационным номером. В аспектах данного изобретения полипептид Dnmt3A кодируется последовательностью нуклеиновой кислоты, идентифицированной по референсной последовательности NCBI с номером доступа NM_022552, по гомологам или по функциональным фрагментам последовательности с данным номером доступа. В аспектах данного изобретения Dnmt3A включает в себя последовательность, изложенную в SEQ ID NO: 26. В аспектах данного изобретения Dnmt3A представляет собой последовательность, изложенную в SEQ ID NO: 26. В аспектах данного изобретения Dnmt3A имеет аминокислотную последовательность, которая по меньшей мере на 50%, на 55%, на 60%, на 65%, на 70%, на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98%, на 99% или на 100% идентична последовательности с SEQ ID NO: 26. В аспектах данного изобретения домен метилтрансферазы ДНК имеет аминокислотную последовательность, которая по меньшей мере на 75% идентична последовательности с SEQ ID NO: 26. В аспектах данного изобретения домен метилтрансферазы ДНК имеет аминокислотную последовательность, которая по меньшей мере на 80% идентична последовательности с SEQ ID NO: 26. В аспектах данного изобретения домен метилтрансферазы ДНК имеет аминокислотную последовательность, которая по меньшей мере на 85% идентична последовательности с SEQ ID NO: 26. В аспектах данного изобретения Dnmt3A имеет аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 26. В аспектах данного изобретения Dnmt3A имеет аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 26.
[0066] Белок «Dnmt3L», «ДНК (цитозин-5)-метилтрансфераза 3L» или «метилтрансфераза ДНК 3L», указанный в данном документе, включает в себя любую из рекомбинантных или встречающихся в природе форм фермента Dnmt3L, либо варианты или гомологи, которые сохраняют активность фермента Dnmt3L (например, в пределах по меньшей мере 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% активности по сравнению с Dnmt3L). В аспектах данного изобретения варианты или гомологи по меньшей мере на 50%, на 55%, на 60%, на 65%, на 70%, на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98%, на 99% или на 100% идентичны аминокислотной последовательности по всей длине последовательности, или на участке последовательности (например, на непрерывном участке длиной 50, 100, 150 или 200 аминокислот) по сравнению с белком Dnmt3L, встречающимся в природе. В аспектах данного изобретения белок Dnmt3L по существу идентичен белку, идентифицированному по регистрационному номеру UniProt Q9CWR8, либо варианту или гомологу, по существу идентичному белку с данным регистрационным номером. В аспектах данного изобретения белок Dnmt3L идентичен белку, идентифицированному по регистрационному номеру UniProt Q9CWR8. В аспектах данного изобретения последовательность белка Dnmt3L по меньшей мере на 75% идентична аминокислотной последовательности белка, идентифицированного по регистрационному номеру UniProt Q9CWR8. В аспектах данного изобретения последовательность белка Dnmt3L по меньшей мере на 80% идентична аминокислотной последовательности белка, идентифицированного по регистрационному номеру UniProt Q9CWR8. В аспектах данного изобретения последовательность белка Dnmt3L по меньшей мере на 85% идентична аминокислотной последовательности белка, идентифицированного по регистрационному номеру UniProt Q9CWR8. В аспектах данного изобретения последовательность белка Dnmt3L по меньшей мере на 95% идентична аминокислотной последовательности белка, идентифицированного по регистрационному номеру UniProt Q9CWR8.
[0067] В аспектах данного изобретения белок Dnmt3L по существу идентичен белку, идентифицированному по регистрационному номеру UniProt Q9UJW, либо варианту или гомологу, по существу идентичному белку с данным регистрационным номером. В аспектах данного изобретения белок Dnmt3L идентичен белку, идентифицированному по регистрационному номеру UniProt Q9UJW. В аспектах данного изобретения последовательность белка Dnmt3L по меньшей мере на 50% идентична аминокислотной последовательности белка, идентифицированного по регистрационному номеру UniProt Q9UJW. В аспектах данного изобретения последовательность белка Dnmt3L по меньшей мере на 55% идентична аминокислотной последовательности белка, идентифицированного по регистрационному номеру UniProt Q9UJW. В аспектах данного изобретения последовательность белка Dnmt3L по меньшей мере на 60% идентична аминокислотной последовательности белка, идентифицированного по регистрационному номеру UniProt Q9UJW. В аспектах данного изобретения последовательность белка Dnmt3L по меньшей мере на 65% идентична аминокислотной последовательности белка, идентифицированного по регистрационному номеру UniProt Q9UJW. В аспектах данного изобретения последовательность белка Dnmt3L по меньшей мере на 70% идентична аминокислотной последовательности белка, идентифицированного по регистрационному номеру UniProt Q9UJW. В аспектах данного изобретения последовательность белка Dnmt3L по меньшей мере на 75% идентична аминокислотной последовательности белка, идентифицированного по регистрационному номеру UniProt Q9UJW. В аспектах данного изобретения последовательность белка Dnmt3L по меньшей мере на 80% идентична аминокислотной последовательности белка, идентифицированного по регистрационному номеру UniProt Q9UJW. В аспектах данного изобретения последовательность белка Dnmt3L по меньшей мере на 85% идентична аминокислотной последовательности белка, идентифицированного по регистрационному номеру UniProt Q9UJW. В аспектах данного изобретения последовательность белка Dnmt3L по меньшей мере на 90% идентична аминокислотной последовательности белка, идентифицированного по регистрационному номеру UniProt Q9UJW. В аспектах данного изобретения последовательность белка Dnmt3L по меньшей мере на 95% идентична аминокислотной последовательности белка, идентифицированного по регистрационному номеру UniProt Q9UJW. В аспектах данного изобретения полипептид Dnmt3L кодируется последовательностью нуклеиновой кислоты, идентифицированной по референсной последовательности NCBI с номером доступа NM_001081695, либо по гомологам или по функциональным фрагментам последовательности с данным номером доступа. В аспектах данного изобретения Dnmt3L включает в себя последовательность, изложенную в SEQ ID NO: 28. В аспектах данного изобретения Dnmt3L представляет собой последовательность, изложенную в SEQ ID NO: 28. В аспектах данного изобретения Dnmt3L имеет аминокислотную последовательность, которая по меньшей мере на 50%, на 55%, на 60%, на 65%, на 70%, на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98%, на 99% или на 100% идентична последовательности с SEQ ID NO: 28. В аспектах данного изобретения Dnmt3L имеет аминокислотную последовательность, которая по меньшей мере на 50% идентична последовательности с SEQ ID NO: 28. В аспектах данного изобретения Dnmt3L имеет аминокислотную последовательность, которая по меньшей мере на 55% идентична последовательности с SEQ ID NO: 28. В аспектах данного изобретения Dnmt3L имеет аминокислотную последовательность, которая по меньшей мере на 60% идентична последовательности с SEQ ID NO: 28. В аспектах данного изобретения Dnmt3L имеет аминокислотную последовательность, которая по меньшей мере на 65% идентична последовательности с SEQ ID NO: 28. В аспектах данного изобретения Dnmt3L имеет аминокислотную последовательность, которая по меньшей мере на 97% идентична последовательности с SEQ ID NO: 28. В аспектах данного изобретения Dnmt3L имеет аминокислотную последовательность, которая по меньшей мере на 75% идентична последовательности с SEQ ID NO: 28. В аспектах данного изобретения Dnmt3L имеет аминокислотную последовательность, которая по меньшей мере на 80% идентична последовательности с SEQ ID NO: 28. В аспектах данного изобретения Dnmt3L имеет аминокислотную последовательность, которая по меньшей мере на 85% идентична последовательности с SEQ ID NO: 28. В аспектах данного изобретения Dnmt3L имеет аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 28. В аспектах данного изобретения Dnmt3L имеет аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 28.
[0068] Термин «РНК-направляемая эндонуклеаза ДНК» и ему подобные термины относятся в обычном и общепринятом смысле к ферменту, который расщепляет фосфодиэфирную связь в полинуклеотидной цепи ДНК, при этом распознаванию фосфодиэфирной связи способствует отдельная последовательность РНК (например, единая гидовая РНК).
[0069] Термин «эндонуклеаза CRISPR класса II» относится к эндонуклеазам, которые обладают такой же эндонуклеазной активностью, как Cas9, и участвуют в системе CRISPR класса II. Примером системы CRISPR класса II является локус CRISPR типа II из Streptococcus pyogenes SF370, который содержит кластер из четырех генов - Cas9, Cas1, Cas2 и Csn1, а также два некодирующих элемента РНК - tracrРНК и характерный массив повторяющихся последовательностей (прямые повторы), разделенных короткими отрезками неповторяющихся последовательностей (спейсеры, около 30 п.о. каждый). Фермент Cpf1 относится к предполагаемой системе CRISPR-Cas типа V. Системы как типа II, так и типа V включены в класс II системы CRISPR-Cas.
[0070] «Последовательность ядерной локализации», или «сигнал ядерной локализации», или «СЯЛ» (англ. «NLS») представляет собой пептид, который направляет белки в ядро. В аспектах данного изобретения СЯЛ включает в себя пять основных положительно заряженных аминокислот. СЯЛ может располагаться в любом месте пептидной цепи. В аспектах данного изобретения СЯЛ представляет собой СЯЛ, полученный от SV40. В аспектах данного изобретения СЯЛ включает в себя последовательность, изложенную в SEQ ID NO: 25. В аспектах данного изобретения СЯЛ представляет собой последовательность, изложенную в SEQ ID NO: 25. В аспектах данного изобретения СЯЛ имеет аминокислотную последовательность, которая по меньшей мере на 50%, на 55%, на 60%, на 65%, на 70%, на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98%, на 99% или на 100% идентична последовательности с SEQ ID NO: 25. В аспектах данного изобретения СЯЛ имеет аминокислотную последовательность, которая по меньшей мере на 75% идентична последовательности с SEQ ID NO: 25. В аспектах данного изобретения СЯЛ имеет аминокислотную последовательность, которая по меньшей мере на 80% идентична последовательности с SEQ ID NO: 25. В аспектах данного изобретения СЯЛ имеет аминокислотную последовательность, которая по меньшей мере на 85% идентична последовательности с SEQ ID NO: 25. В аспектах данного изобретения СЯЛ имеет аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 25. В аспектах данного изобретения СЯЛ имеет аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 25. В аспектах данного изобретения СЯЛ имеет аминокислотную последовательность с SEQ ID NO: 25.
[0071] Термин «клетка», употребляемый в контексте данного документа, относится к клетке, выполняющей метаболическую или другую функцию, достаточную для сохранения или репликации ее геномной ДНК. Клетка может быть идентифицирована с помощью методов, которые хорошо известны в данной области техники, включая, например, наличие неповрежденной мембраны, окрашивание определенным красителем, способность производить потомство или, в случае гаметы, способность объединяться со второй гаметой для производства жизнеспособного потомства. Клетки могут включать в себя прокариотические и эукариотические клетки. Прокариотические клетки включают в себя, но не ограничиваются ими, бактерии. Эукариотические клетки включают в себя, но не ограничиваются ими, дрожжевые клетки и клетки, полученные из растений и животных, например, клетки млекопитающих, насекомых (например, Spodoptera) и человека. Клетки могут быть применимы, когда они от природы являются неприлипающими, или были обработаны так, чтобы не прилипать к поверхностям, например, путем трипсинизации.
[0072] Термин «вектор», употребляемый в контексте данного документа, относится к молекуле нуклеиновой кислоты, способной переносить другую нуклеиновую кислоту, с которой она была связана. Одним из типов вектора является «плазмида», которая относится к линейной или кольцевой двухцепочечной петле ДНК, в которую могут быть лигированы дополнительные сегменты ДНК. Другой тип вектора представляет собой вирусный вектор, в котором дополнительные сегменты ДНК могут быть лигированы в вирусный геном. Определенные векторы способны к автономной репликации в клетке-хозяине, в которую они введены (например, бактериальные векторы, имеющие бактериальную точку начала репликации, и эписомальные векторы млекопитающих). Другие векторы (например, неэписомные векторы млекопитающих) интегрируются в геном клетки-хозяина после введения в клетку-хозяина и, таким образом, реплицируются вместе с геномом хозяина. Более того, определенные векторы способны управлять экспрессией генов, с которыми они функционально связаны. Такие векторы именуются в данном документе «векторами экспрессии». В общем, векторы экспрессии, используемые в технологиях рекомбинантных ДНК, часто находятся в форме плазмид. В данном документе термины «плазмида» и «вектор» могут употребляться взаимозаменяемо, поскольку плазмида является наиболее часто используемой формой вектора. Тем не менее, данное изобретение предназначено для включения других форм векторов экспрессии, таких как вирусные векторы (например, ретровирусы с дефектом репликации, аденовирусы и аденоассоциированные вирусы), которые выполняют эквивалентные функции. Кроме того, некоторые вирусные векторы способны нацеливаться на конкретный тип клеток либо специфично, либо неспецифично. Неспособные к репликации вирусные векторы, или вирусные векторы с дефектом репликации, относятся к вирусным векторам, которые способны инфицировать свои клетки-мишени и доставлять свою вирусную нагрузку, но затем не могут продолжить типичный литический путь, который приводит к лизису и гибели клеток.
[0073] Термины «трансфекция», «трансдукция», «трансфицировать» или «трансдуцировать» могут употребляться взаимозаменяемо, и определяются как процесс введения молекулы нуклеиновой кислоты и (или) белка в клетку. Нуклеиновые кислоты могут быть введены в клетку с использованием невирусных методов или методов, основанных на вирусах. Молекула нуклеиновой кислоты может представлять собой последовательность, кодирующую полные белки или их функциональные части. Обычно вектор нуклеиновой кислоты содержит элементы, необходимые для экспрессии белка (например, промотор, сайт начала транскрипции и т. д.). Невирусные методы трансфекции включают в себя любой подходящий метод, в котором не используется вирусная ДНК или вирусные частицы в качестве системы доставки для введения молекулы нуклеиновой кислоты в клетку. Типичные методы невирусной трансфекции включают в себя инкапсуляцию наночастиц нуклеиновых кислот, которые кодируют слитый белок (например, липидные наночастицы, наночастицы золота и тому подобное), трансфекцию фосфатом кальция, липосомную трансфекцию, нуклеофекцию, сонопорацию, трансфекцию посредством теплового шока, трансфекцию посредством намагничивания и электропорацию. Что касается методов, основанных на вирусах, то любой пригодный вирусный вектор может быть использован в способах, описанных в данном документе. Примеры вирусных векторов включают, но не ограничиваются ими, ретровирусные, аденовирусные, лентивирусные и аденоассоциированные вирусные векторы. В аспектах данного изобретения молекулы нуклеиновой кислоты вводятся в клетку с использованием ретровирусного вектора в соответствии со стандартными процедурами, хорошо известными в данной области техники. Термины «трансфекция» или «трансдукция» также относятся к введению белков в клетку из внешней среды. Обычно трансдукция или трансфекция белка основывается на присоединении пептида или белка, способного пересекать клеточную мембрану, к представляющему интерес белку. См., например, Ford et al. (2001) Gene Therapy 8: 1-4, и Prochiantz (2007) Nat. Methods 4: 119-20.
[0074] Термин «пептидный линкер», употребляемый в контексте данного документа, представляет собой линкер, включающий в себя пептидный фрагмент. В вариантах реализации данного изобретения пептидный линкер представляет собой двухвалентный пептид, такой как аминокислотная последовательность, присоединенная на N-конце и C-конце к остатку соединения (например, слитый белок, представленный в данном документе). Пептидный линкер может быть пептидным фрагментом (двухвалентным пептидным фрагментом), который можно расщепить (например, полипептидом, расщепляемым P2A). Пептидный линкер в контексте данного документа также может взаимозаменяемо именоваться аминокислотным линкером. В аспектах данного изобретения пептидный линкер включает в себя от 1 до около 80 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя от 1 до около 70 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя от 1 до около 60 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя от 1 до около 50 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя от 1 до около 40 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя от 1 до около 30 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя от 1 до около 25 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя от 1 до около 20 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя от около 2 до около 20 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя от около 2 до около 19 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя от около 2 до около 18 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя от около 2 до около 17 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя от около 2 до около 16 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя от около 2 до около 15 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя от около 2 до около 14 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя от около 2 до около 13 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя от около 2 до около 12 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя от около 2 до около 11 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя от около 2 до около 10 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя от около 2 до около 9 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя от около 2 до около 8 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя от около 2 до около 7 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя от около 2 до около 6 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя от около 2 до около 5 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя от около 2 до около 4 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя от около 2 до около 3 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя от около 3 до около 19 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя от около 3 до около 18 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя от около 3 до около 17 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя от около 3 до около 16 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя от около 3 до около 15 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя от около 3 до около 14 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя от около 3 до около 13 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя от около 3 до около 12 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя от около 3 до около 11 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя от около 3 до около 10 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя от около 3 до около 9 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя от около 3 до около 8 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя от около 3 до около 7 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя от около 3 до около 6 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя от около 3 до около 5 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя от около 3 до около 4 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя от около 10 до около 20 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя от около 15 до около 20 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя около 2 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя около 3 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя около 4 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя около 5 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя около 6 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя около 7 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя около 8 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя около 9 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя около 10 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя около 11 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя около 12 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя около 13 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя около 14 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя около 15 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя около 16 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя около 17 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя около 18 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя около 19 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя около 20 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя около 21 аминокислотного остатка. В аспектах данного изобретения пептидный линкер включает в себя около 22 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя около 23 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя около 24 аминокислотных остатков. В аспектах данного изобретения пептидный линкер включает в себя около 25 аминокислотных остатков.
[0075] В аспектах данного изобретения пептидный линкер включает в себя последовательность, изложенную в SEQ ID NO: 17. В аспектах данного изобретения пептидный линкер представляет собой последовательность, изложенную в SEQ ID NO: 17. В аспектах данного изобретения пептидный линкер включает в себя последовательность, изложенную в SEQ ID NO: 18. В аспектах данного изобретения пептидный линкер представляет собой последовательность, изложенную в SEQ ID NO: 18. В аспектах данного изобретения пептидный линкер включает в себя последовательность, изложенную в SEQ ID NO: 19. В аспектах данного изобретения пептидный линкер представляет собой последовательность, изложенную в SEQ ID NO: 19. В аспектах данного изобретения пептидный линкер включает в себя последовательность, изложенную в SEQ ID NO: 20. В аспектах данного изобретения пептидный линкер представляет собой последовательность, изложенную в SEQ ID NO: 20. В аспектах данного изобретения пептидный линкер включает в себя последовательность, изложенную в SEQ ID NO: 21. В аспектах данного изобретения пептидный линкер представляет собой последовательность, изложенную в SEQ ID NO: 21. В аспектах данного изобретения пептидный линкер включает в себя последовательность, изложенную в SEQ ID NO: 22. В аспектах данного изобретения пептидный линкер представляет собой последовательность, изложенную в SEQ ID NO: 22. В аспектах данного изобретения пептидный линкер включает в себя последовательность, изложенную в SEQ ID NO: 27. В аспектах данного изобретения пептидный линкер представляет собой последовательность, изложенную в SEQ ID NO: 27. В аспектах данного изобретения пептидный линкер включает в себя последовательность, изложенную в SEQ ID NO: 24. В аспектах данного изобретения пептидный линкер представляет собой последовательность, изложенную в SEQ ID NO: 24. В аспектах данного изобретения пептидный линкер включает в себя последовательность, изложенную в SEQ ID NO: 29. В аспектах данного изобретения пептидный линкер представляет собой последовательность, изложенную в SEQ ID NO: 29. В аспектах данного изобретения пептидный линкер представляет собой полипептид XTEN. В аспектах данного изобретения пептидный линкер имеет аминокислотную последовательность, которая по меньшей мере на 50%, на 55%, на 60%, на 65%, на 70%, на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98%, на 99% или на 100% идентична последовательности с SEQ ID NO: 17, 18, 19, 20, 21, 22, 24, 27 или 29. В аспектах данного изобретения пептидный линкер имеет аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 17, 18, 19, 20, 21, 22, 24, 27 или 29.
[0076] В аспектах данного изобретения пептидный линкер имеет аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности SEQ ID NO: 17. В аспектах данного изобретения пептидный линкер имеет аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 18. В аспектах данного изобретения пептидный линкер имеет аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 19. В аспектах данного изобретения пептидный линкер имеет аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 20. В аспектах данного изобретения пептидный линкер имеет аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 21. В аспектах данного изобретения пептидный линкер имеет аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 22. В аспектах данного изобретения пептидный линкер имеет аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 24. В аспектах данного изобретения пептидный линкер имеет аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 27. В аспектах данного изобретения пептидный линкер имеет аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 29. В аспектах данного изобретения пептидный линкер имеет аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 17, 18, 19, 20, 21, 22, 24, 27 или 29. В аспектах данного изобретения пептидный линкер имеет аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 17. В аспектах данного изобретения пептидный линкер имеет аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 18. В аспектах данного изобретения пептидный линкер имеет аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 19. В аспектах данного изобретения пептидный линкер имеет аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 20. В аспектах данного изобретения пептидный линкер имеет аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 21. В аспектах данного изобретения пептидный линкер имеет аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 22. В аспектах данного изобретения пептидный линкер имеет аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 24. В аспектах данного изобретения пептидный линкер имеет аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 27. В аспектах данного изобретения пептидный линкер имеет аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 29.
[0077] Термины «XTEN», «линкер XTEN» или «полипептид XTEN» в контексте данного документа относятся к рекомбинантному полипептиду (например, неструктурированному рекомбинантному пептиду), не имеющему гидрофобных аминокислотных остатков. О разработке и использовании XTEN можно найти, например, в публикации Schellenberger et al., Nature Biotechnology 27, 1186-1190 (2009), которая полностью и для всех целей включена в данный документ посредством ссылки. В аспектах данного изобретения линкер XTEN включает в себя последовательность, изложенную в SEQ ID NO: 31. В аспектах данного изобретения линкер XTEN представляет собой последовательность, изложенную в SEQ ID NO: 31. В аспектах данного изобретения линкер XTEN включает в себя последовательность, изложенную SEQ ID NO: 32. В аспектах данного изобретения линкер XTEN представляет собой последовательность, изложенную в SEQ ID NO: 32. В аспектах данного изобретения линкер XTEN имеет аминокислотную последовательность, которая по меньшей мере на 50%, на 55%, на 60%, на 65%, на 70%, на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98%, на 99% или на 100% идентична последовательности с SEQ ID NO: 31. В аспектах данного изобретения линкер XTEN имеет аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 31. В аспектах данного изобретения линкер XTEN имеет аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 31. В аспектах данного изобретения линкер XTEN имеет аминокислотную последовательность, которая по меньшей мере на 50%, на 55%, на 60%, на 65%, на 70%, на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98%, на 99% или на 100% идентична последовательности с SEQ ID NO: 32. В аспектах данного изобретения линкер XTEN имеет аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 32. В аспектах данного изобретения линкер XTEN имеет аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 32.
[0078] «Детектируемый агент» или «детектируемый фрагмент» представляет собой композицию, которую можно детектировать соответствующими средствами, такими как спектроскопические, фотохимические, биохимические, иммунохимические, химические средства, магнитно-резонансная томография или другие физические средства. Например, пригодные реагенты для детекции включают в себя 18F, 32P, 33P, 45Ti, 47Sc, 52Fe, 59Fe, 62Cu, 64Cu, 67Cu, 67Ga, 68Ga, 77As, 86Y, 90Y. 89Sr, 89Zr, 94Tc, 94Tc, 99mTc, 99Mo, 105Pd, 105Rh, 111Ag, 111In, 123I, 124I, 125I, 131I, 142Pr, 143Pr, 149Pm, 153Sm, 154-1581Gd, 161Tb, 166Dy, 166Ho, 169Er, 175Lu, 177Lu, 186Re, 188Re, 189Re, 194Ir, 198Au, 199Au, 211At, 211Pb, 212Bi, 212Pb, 213Bi, 223Ra, 225Ac, Cr, V, Mn, Fe, Co, Ni, Cu, La, Ce, Pr, Nd, Pm, Sm, Eu, Gd, Tb, Dy, Ho, Er, Tm, Yb, Lu, 32P, флуорофор (например, флуоресцентные красители), электронно-плотные реагенты, ферменты (например, широко используемые в твердофазном ИФА), биотин, дигоксигенин, парамагнитные молекулы, парамагнитные наночастицы, сверхмалые суперпарамагнитные наночастицы оксида железа (англ. «USPIO»), агрегаты наночастиц USPIO, суперпарамагнитные наночастицы оксида железа (англ. «SPIO»), агрегаты наночастиц SPIO, монокристаллические наночастицы оксида железа, монокристаллический оксид железа, контрастные вещества на основе наночастиц, липосомы или другие средства доставки, содержащие молекулы хелата гадолиния («хелат Gd») молекулы, гадолиний, радиоизотопы, радионуклиды (например, углерод-11, азот-13, кислород-15, фтор-18, рубидий-82), фтордезоксиглюкоза (например, меченная фтором-18), любые излучающие гамма-лучи радионуклиды, излучающие позитроны радионуклиды, радиоактивно меченая глюкоза, радиоактивно меченная вода, радиоактивно меченый аммиак, биоколлоиды, микропузырьки (например, включительно с оболочками микропузырьков, содержащими альбумин, галактозу, липиды и (или) полимеры; включительно с газовым ядром микропузырьков, содержащим воздух, тяжелый газ (тяжелые газы), перфторуглерод, азот, октафторпропан, перфлексановые липидные микросферы, перфлутрен и т. д.), йодированные контрастные вещества (например, иогексол, йодиксанол, иоверсол, иопамидол, иоксилан, иопромид, диатризоат, метризоат, иоксаглат), сульфат бария, диоксид тория, золото, золотые наночастицы, агрегаты золотых наночастиц, флуорофоры, двухфотонные флуорофоры или гаптены и белки, или другие объекты, которые могут быть детектированы, например, путем включения радиоактивной метки в пептид или антитело, специфически реагирующие с целевым пептидом.
[0079] Детектируемый фрагмент представляет собой одновалентный детектируемый агент или детектируемый агент, способный образовывать связь с другой композицией. В аспектах данного изобретения детектируемый агент представляет собой метку HA. В аспектах данного изобретения метка HA включает в себя последовательность, изложенную в SEQ ID NO: 24. В аспектах данного изобретения метка НА представляет собой последовательность, изложенную в SEQ ID NO: 24. В аспектах данного изобретения метка НА имеет аминокислотную последовательность, которая по меньшей мере на 80% идентична последовательности с SEQ ID NO: 24. В аспектах данного изобретения метка НА имеет аминокислотную последовательность, которая по меньшей мере на 85% идентична последовательности с SEQ ID NO: 24. В аспектах данного изобретения метка НА имеет аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 24. В аспектах данного изобретения метка НА имеет аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 24. В аспектах данного изобретения детектируемый агент представляет собой синий флуоресцентный белок (СФБ, англ. «BFP»). В аспектах данного изобретения СФБ включает в себя последовательность, изложенную в SEQ ID NO: 30. В аспектах данного изобретения СФБ представляет собой последовательность, изложенную в SEQ ID NO: 30. В аспектах данного изобретения СФБ имеет аминокислотную последовательность, которая по меньшей мере на 80% идентична последовательности с SEQ ID NO: 30. В аспектах данного изобретения СФБ имеет аминокислотную последовательность, которая по меньшей мере на 85% идентична последовательности с SEQ ID NO: 30. В аспектах данного изобретения СФБ имеет аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 30. В аспектах данного изобретения СФБ имеет аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 30.
[0080] Радиоактивные вещества (например, радиоизотопы), которые можно использовать в качестве агентов для визуализации и (или) маркировки в соответствии с аспектами данного изобретения, включают в себя, но не ограничиваются ими, 18F, 32P, 33P, 45Ti, 47Sc, 52Fe, 59Fe, 62Cu, 64Cu, 67Cu, 67Ga, 68Ga, 77As, 86Y, 90Y, 89Sr, 89Zr, 94Tc, 94Tc, 99mTc, 99Mo, 105Pd, 105Rh, 111Ag, 111In, 123I, 124I, 125I, 131I, 142Pr, 143Pr, 149Pm, 153Sm, 154-1581Gd, 161Tb, 166Dy, 166Ho, 169Er, 175Lu, 177Lu, 186Re, 188Re, 189Re, 194Ir, 198Au, 199Au, 211At, 211Pb, 212Bi, 212Pb, 213Bi, 223Ra и 225Ac. Парамагнитные ионы, которые могут использоваться в качестве дополнительных агентов для визуализации в соответствии с аспектами данного изобретения, включают в себя, но не ограничиваются ими, ионы переходных металлов и лантаноидных металлов (например, металлы с атомными номерами 21-29, 42, 43, 44 или 57-71). Эти металлы включают в себя ионы Cr, V, Mn, Fe, Co, Ni, Cu, La, Ce, Pr, Nd, Pm, Sm, Eu, Gd, Tb, Dy, Ho, Er, Tm, Yb и Lu.
[0081] Термин «контактирование» употребляется в его простом, обычном значении и относится к процессу, позволяющему по крайней мере двум различным молекулам приблизиться друг к другу на достаточно близкое расстояние, чтобы реагировать, взаимодействовать или физически соприкасаться. Однако следует понимать, что конечный продукт реакции может быть получен непосредственно в результате реакции между добавленными реагентами, или из промежуточного продукта из одного или нескольких добавленных реагентов, которые могут быть получены в реакционной смеси.
[0082] Термин «контактирование» может включать в себя предоставление возможности двум молекулам реагировать, взаимодействовать или физически соприкасаться, где две молекулы могут представлять собой, например, слитый белок, как предусмотрено в данном документе, и последовательность нуклеиновой кислоты (например, последовательность целевой ДНК).
[0083] Как определено в данном документе, термин «ингибирование», «ингибирует», «ингибирующий», «репрессия», «репрессирует», «сайленсинг», «вызывать сайленсинг» и тому подобное при употреблении в отношении композиции, представленной в данном документе (например, слитого белка, комплекса, нуклеиновой кислоты, вектора), относятся к отрицательному влиянию (например, снижению) активности (например, транскрипции) последовательности нуклеиновой кислоты (например, снижению транскрипции гена) относительно активности последовательности нуклеиновой кислоты (например, транскрипции гена) в отсутствие композиции (например, слитого белка, комплекса, нуклеиновой кислоты, вектора). В аспектах данного изобретения ингибирование относится к ослаблению заболевания или симптомов заболевания (например, онкологического заболевания). Таким образом, ингибирование включает, по крайней мере частично, частичное или полное блокирование активности (например, транскрипции) или уменьшение, предотвращение или задержку активности (например, транскрипции) последовательности нуклеиновой кислоты. Ингибированная активность (например, транскрипция) может составлять 90%, 80%, 70%, 60%, 50%, 40%, 30%, 20%, 10% или меньше, чем активность в контроле. В аспектах данного изобретения ингибирование является 1,5-кратным, 2-кратным, 3-кратным, 4-кратным, 5-кратным, 10-кратным или более по сравнению с контролем.
[0084] «Контрольный» образец или значение относится к образцу, который служит референсом, обычно известным референсом, для сравнения с исследуемым образцом. Например, исследуемый образец может быть отобран из исследуемого условия, например, в присутствии исследуемого соединения, и сравнен с образцами из известных условий, например, в отсутствие исследуемого соединения (отрицательный контроль) или в присутствии известного соединения (положительный контроль). Контроль также может представлять собой среднее значение, полученное из ряда исследований или результатов. Специалист в данной области техники поймет, что контроли могут быть разработаны для оценки любого количества параметров. Например, можно разработать контроль для сравнения терапевтического эффекта на основе фармакологических данных (например, период полужизни) или терапевтических мер (например, сравнение побочных эффектов). Специалист в данной области техники поймет, какие контроли важны в определенной ситуации, и сможет анализировать данные на основе сравнений с контрольными значениями. Контроли также важны для определения значимости данных. Например, если значения для определенного параметра сильно различаются в контроле, то вариация в исследуемых образцах не будет считаться значимой.
[0085] Слитые белки
[0086] В данном документе представлены, среди прочего, слитые белки, которые могут отключать гены навсегда (например, необратимо) и обратимо в клетках млекопитающих с использованием редактирования эпигенома на основе CRISPR. В вариантах реализации данного изобретения слитый белок включает в себя один полипептид, слитый из четырех белков (например, каталитически неактивного Cas9 (например, dCas9), домена KRAB, Dnmt3A и Dnmt3L), которые могут кратковременно экспрессироваться в клетках. Слитый белок может быть направлен в конкретный сайт в геноме млекопитающего с использованием полинуклеотида, комплементарного целевой последовательности нуклеиновой кислоты (например, последовательности ДНК), и который дополнительно включает в себя последовательность (т. е. связывающую последовательность), способную к связыванию со слитым белком. После правильного позиционирования, и без намерения быть связанным теорией, слитый белок добавляет маркеры метилирования ДНК и (или) репрессивные хроматиновые маркеры к целевой нуклеиновой кислоте, что приводит к сайленсингу гена, который наследуется при последующих делениях клеток. Таким образом, слитый белок может выполнять редактирование эпигенома, которое позволяет обойтись без создания двухцепочечных разрывов ДНК в геноме хозяина, что делает его безопасным и обратимым способом манипулирования геномом живого организма.
[0087] В вариантах реализации данного изобретения слитый белок содержит фермент нуклеазо-дефицитную РНК-направляемую эндонуклеазу ДНК; домен KRAB и домен метилтрансферазы ДНК. В аспектах данного изобретения слитый белок содержит, в направлении от N-конца до C-конца, домен метилтрансферазы ДНК, фермент нуклеазо-дефицитную РНК-направляемую эндонуклеазу ДНК, и домен KRAB. В аспектах данного изобретения слитый белок содержит, в направлении от N-конца до C-конца, фермент нуклеазо-дефицитную РНК-направляемую эндонуклеазу ДНК, и домен метилтрансферазы ДНК. В вариантах реализации данного изобретения слитый белок дополнительно содержит один или несколько пептидных линкеров. В аспектах данного изобретения слитый белок дополнительно содержит одну или несколько детектируемых меток. В аспектах данного изобретения слитый белок дополнительно содержит одну или несколько последовательностей ядерной локализации. В аспектах данного изобретения слитый белок дополнительно содержит один или несколько пептидных линкеров, одну или несколько детектируемых меток, одну или несколько последовательностей ядерной локализации, или комбинацию двух или более из вышеперечисленных. Когда слитый белок содержит один или несколько пептидных линкеров, каждый из пептидных лайнеров может быть одинаковым или разными. Когда слитый белок содержит одну или несколько детектируемых меток, каждая из детектируемых меток может быть одинаковой или разной. В аспектах данного изобретения слитый белок содержит от 1 до 10 детектируемых меток. В аспектах данного изобретения слитый белок содержит от 1 до 9 детектируемых меток. В аспектах данного изобретения слитый белок содержит от 1 до 8 детектируемых меток. В аспектах данного изобретения слитый белок содержит от 1 до 7 детектируемых меток. В аспектах данного изобретения слитый белок содержит от 1 до 6 детектируемых меток. В аспектах данного изобретения слитый белок содержит от 1 до 5 детектируемых меток. В аспектах данного изобретения слитый белок содержит от 1 до 4 детектируемых меток. В аспектах данного изобретения слитый белок содержит от 1 до 3 детектируемых меток. В аспектах данного изобретения слитый белок содержит от 1 до 2 детектируемых меток. В аспектах данного изобретения слитый белок содержит 1 детектируемую метку. В аспектах данного изобретения слитый белок содержит 2 детектируемых метки. В аспектах данного изобретения слитый белок содержит 3 детектируемых метки. В аспектах данного изобретения слитый белок содержит 4 детектируемых метки. В аспектах данного изобретения слитый белок содержит 5 детектируемых меток.
[0088] В вариантах реализации данного изобретения слитый белок содержит структуру A - B - C, или B - A - C, или C - A - B, или C - B - A, или B - C - A, или A - C - B; где А представляет собой фермент нуклеазо-дефицитную РНК-направляемую эндонуклеазу ДНК; B представляет собой домен KRAB, C представляет собой домен метилтрансферазы ДНК; и при этом компонент слева является N-концом, а компонент справа является C-концом. В аспектах данного изобретения слитый белок дополнительно содержит один или несколько пептидных линкеров и одну или несколько детектируемых меток. В аспектах данного изобретения каждый из A - B, B - A, B - C, C - B, A - C и C - A независимо связан друг с другом ковалентной связью, пептидным линкером, детектируемой меткой, последовательностью ядерной локализации или комбинацией двух или более из них. Пептидный линкер может быть любым известным в данной области техники (например, пептид, расщепляемый P2A, линкер XTEN и тому подобное). В аспектах данного изобретения слитый белок содержит другие компоненты, такие как детектируемые метки (например, метка НА, синий флуоресцентный белок и т.п.).
[0089] В вариантах реализации данного изобретения слитый белок содержит структуру A - L1 - B - L2 - C, где A представляет собой фермент нуклеазо-дефицитную РНК-направляемую эндонуклеазу ДНК; B представляет собой домен KRAB, C представляет собой домен метилтрансферазы ДНК, L1 представляет собой ковалентную связь или пептидный линкер, и L2 представляет собой ковалентную связь или пептидный линкер; и где A находится на N-конце, а C находится на C-конце. В аспектах данного изобретения A ковалентно связан с B через пептидный линкер. В аспектах данного изобретения A ковалентно связан с B через ковалентную связь. В аспектах данного изобретения B ковалентно связан с C через пептидный линкер. В аспектах данного изобретения B ковалентно связан с C через ковалентную связь. Пептидный линкер может быть любым известным в данной области техники (например, пептид, расщепляемый P2A, линкер XTEN и тому подобное). В аспектах данного изобретения слитый белок содержит другие компоненты, такие как детектируемые метки, последовательности ядерной локализации и т.п. В аспектах данного изобретения L1 представляет собой ковалентную связь, пептидный линкер, детектируемую метку, последовательность ядерной локализации или их комбинацию. В аспектах данного изобретения L2 представляет собой ковалентную связь, пептидный линкер, детектируемую метку, последовательность ядерной локализации или их комбинацию.
[0090] В вариантах реализации данного изобретения слитый белок содержит структуру B - L1 - A - L2 - C, где A представляет собой фермент нуклеазо-дефицитную РНК-направляемую эндонуклеазу ДНК; B представляет собой домен KRAB, C представляет собой домен метилтрансферазы ДНК, L1 представляет собой ковалентную связь или пептидный линкер, и L2 представляет собой ковалентную связь или пептидный линкер; и где B находится на N-конце, а C находится на C-конце. В аспектах данного изобретения L1 представляет собой пептидный линкер. В аспектах данного изобретения L1 представляет собой ковалентную связь. В аспектах данного изобретения L2 представляет собой пептидный линкер. В аспектах данного изобретения L2 представляет собой ковалентную связь. Пептидный линкер может быть любым известным в данной области техники или описанным в данном документе (например, пептид, расщепляемый P2A, линкер XTEN и т.п.). В аспектах данного изобретения слитый белок содержит другие компоненты, такие как детектируемые метки. В аспектах данного изобретения L1 представляет собой ковалентную связь, пептидный линкер, детектируемую метку или их комбинацию. В аспектах данного изобретения L2 представляет собой ковалентную связь, пептидный линкер, детектируемую метку или их комбинацию. В аспектах данного изобретения слитый белок дополнительно содержит последовательность ядерной локализации. Иллюстративные слитые белки, содержащие структуру B - L1 - A - L2 - C, включают в себя p76, p90, p91, p92, p93, p94, p95, p96, p97, p98, p99, p100, p101 и p102 (Фиг. 5)
[0091] В вариантах реализации данного изобретения слитый белок содержит структуру B - L3 - A - L4 - C - L5 - D; где A представляет собой фермент нуклеазо-дефицитную РНК-направляемую эндонуклеазу ДНК; B представляет собой домен KRAB, C представляет собой домен метилтрансферазы ДНК, D отсутствует или D представляет собой одну или несколько детектируемых меток, L3 представляет собой ковалентную связь, пептидный линкер, детектируемую метку или комбинацию двух или более из них, L4 представляет собой ковалентную связь, пептидный линкер, детектируемую метку или комбинацию двух или более из них, L5 отсутствует или L5 представляет собой ковалентную связь или пептидный линкер; и где B находится на N-конце, а D находится на C-конце. В аспектах данного изобретения L3 представляет собой пептидный линкер. В аспектах данного изобретения L3 представляет собой ковалентную связь. В аспектах данного изобретения L3 содержит пептидный линкер и детектируемую метку. В аспектах данного изобретения L3 содержит детектируемую метку. В аспектах данного изобретения L4 представляет собой пептидный линкер. В аспектах данного изобретения L4 содержит пептидный линкер и детектируемую метку. В аспектах данного изобретения L4 представляет собой ковалентную связь. В аспектах данного изобретения L4 содержит обнаруживаемую метку. В аспектах данного изобретения L5 представляет собой пептидный линкер. В аспектах данного изобретения L5 представляет собой ковалентную связь. В аспектах данного изобретения D содержит один или множество детектируемых меток. В аспектах данного изобретения D содержит одну детектируемую метку. В аспектах данного изобретения D содержит две детектируемых метки. В аспектах данного изобретения D содержит три детектируемых метки. В аспектах данного изобретения D содержит множество детектируемых меток. D может быть любой детектируемой меткой, известной в данной области техники и (или) описанной в данной документе (например, меткой НА, синим флуоресцентным белком и т.п.). В аспектах данного изобретения L5 и D могут отсутствовать. Когда L3, L4, L5 и D содержат две или более детектируемых метки, каждая из детектируемых меток является одинаковой или разной. Пептидный линкер может быть любым, известным в данной области техники и (или) описанным в данном документе (например, пептид, расщепляемый P2A, линкер XTEN и т.п.). В аспектах данного изобретения слитый белок дополнительно содержит последовательность ядерной локализации. Иллюстративные слитые белки, содержащие структуру B - L3 - A - L4 - C - L5 - D, включают в себя p76, p90, p91, p92, p93, p94, p95, p96, p97, p98, p99, p100, p101 и p102, как показано на Фиг. 5.
[0092] В вариантах реализации данного изобретения слитый белок содержит структуру C - L3 - A - L4 - B - L5 - D, где A представляет собой фермент нуклеазо-дефицитную РНК-направляемую эндонуклеазу ДНК; B представляет собой домен KRAB, C представляет собой домен метилтрансферазы ДНК, D отсутствует или D содержит одну или несколько детектируемых меток, L3 представляет собой ковалентную связь, пептидный линкер, детектируемую метку или комбинацию двух или более из них, L4 представляет собой ковалентную связь, пептидный линкер, детектируемую метку или комбинацию двух или более из них, L5 отсутствует или L5 представляет собой ковалентную связь или пептидный линкер; и где C находится на N-конце, а D находится на C-конце. В аспектах данного изобретения L3 представляет собой пептидный линкер. В аспектах данного изобретения L3 представляет собой ковалентную связь. В аспектах данного изобретения L3 содержит детектируемую метку. В аспектах данного изобретения L3 содержит пептидный линкер и детектируемую метку. В аспектах данного изобретения L4 представляет собой пептидный линкер. В аспектах данного изобретения L4 представляет собой ковалентную связь. В аспектах данного изобретения L4 содержит обнаруживаемую метку. В аспектах данного изобретения L4 содержит пептидный линкер и детектируемую метку. В аспектах данного изобретения L5 представляет собой пептидный линкер. В аспектах данного изобретения L5 представляет собой ковалентную связь. В аспектах данного изобретения D содержит один или множество детектируемых меток. В аспектах данного изобретения D содержит одну детектируемую метку. В аспектах данного изобретения D содержит две детектируемых метки. В аспектах данного изобретения D содержит три детектируемых метки. В аспектах данного изобретения D содержит множество детектируемых меток. D может быть любой детектируемой меткой, известной в данной области техники и (или) описанной в данной документе (например, меткой НА, синим флуоресцентным белком и т.п.). В аспектах данного изобретения L5 и D могут отсутствовать. Когда L3, L4, L5 и D содержат две или более детектируемых метки, каждая из детектируемых меток является одинаковой или разной. Пептидный линкер может быть любым, известным в данной области техники и (или) описанным в данном документе (например, пептид, расщепляемый P2A, линкер XTEN и т.п.). В аспектах данного изобретения слитый белок дополнительно содержит последовательность ядерной локализации. Иллюстративные слитые белки, содержащие структуру C - L3 - A - L4 - B - L5 - D, включают в себя p112, как показано на Фиг. 5.
[0093] Термин «фермент нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК» и тому подобные относятся в обычном и общепринятом смысле к РНК-направляемой эндонуклеазе ДНК (например, к мутантной форме встречающейся в природе РНК-направляемой эндонуклеазы ДНК), которая нацелена на специфическую фосфодиэфирную связь в полинуклеотиде ДНК, при этом распознаванию фосфодиэфирной связи способствует отдельная полинуклеотидная последовательность (например, последовательность РНК (например, единая гидовая РНК (егРНК)), но которая не способна расщеплять целевую фосфодиэфирную связь до значительной степени (например, отсутствует измеримый разрыв фосфодиэфирной связи в физиологических условиях). Нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК, таким образом, сохраняет ДНК-связывающую способность (например, специфическое связывание с целевой последовательностью) при образовании комплекса с полинуклеотидом (например, егРНК), но не обладает значительной эндонуклеазной активностью (например, каким-либо детектируемым уровнем эндонуклеазной активности). В аспектах данного изобретения фермент нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК представляет собой dCas9, ddCpf1, нуклеазо-дефицитный вариант Cas9, или нуклеазо-дефицитную эндонуклеазу CRISPR класса II.
[0094] В вариантах реализации данного изобретения фермент нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК представляет собой dCas9. Термины «dCas9» или «белок dCas9» в контексте данного документа относятся к белку Cas9, в котором оба каталитических сайта для эндонуклеазной активности являются дефектными или неактивными. В аспектах данного изобретения белок dCas9 имеет мутации в положениях, соответствующих D10A и H840A в Cas9 от S. pyogenes. В аспектах данного изобретения белок dCas9 лишен эндонуклеазной активности из-за точечных мутаций в обоих каталитических эндонуклеазных сайтах (RuvC и HNH) Cas9 дикого типа. Точечные мутации могут представлять собой D10A и H840A. В аспектах данного изобретения dCas9 по существу не обладает детектируемой эндонуклеазной (например, эндодезоксирибонуклеазной) активностью. В аспектах данного изобретения dCas9 включает в себя аминокислотную последовательность с SEQ ID NO: 23. В аспектах данного изобретения dCas9 имеет аминокислотную последовательность с SEQ ID NO: 23. В аспектах данного изобретения dCas9 имеет аминокислотную последовательность, которая по меньшей мере на 50%, на 55%, на 60%, на 65%, на 70%, на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98%, на 99% или на 100% идентична последовательности с SEQ ID NO: 23. В аспектах данного изобретения dCas9 имеет аминокислотную последовательность, которая по меньшей мере на 75% идентична последовательности с SEQ ID NO: 23. В аспектах данного изобретения dCas9 имеет аминокислотную последовательность, которая по меньшей мере на 80% идентична последовательности с SEQ ID NO: 23. В аспектах данного изобретения dCas9 имеет аминокислотную последовательность, которая по меньшей мере на 85% идентична последовательности с SEQ ID NO: 23. В аспектах данного изобретения dCas9 имеет аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 23. В аспектах данного изобретения dCas9 имеет аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 23.
[0095] Термины «связанный с CRISPR белок 9», «Cas9», «Csn1» или «белок Cas9» при употреблении в контексте данного документа включают в себя любую из рекомбинантных или встречающихся в природе форм эндонуклеазы Cas9, либо их варианты или гомологи, которые сохраняют эндонуклеазную активность фермента Cas9 (например, в пределах по меньшей мере 50%, 80%, 90%, 95%, 96%, 97%, 98%, 99% или 100% активности по сравнению с Cas9). В некоторых аспектах данного изобретения варианты или гомологи по меньшей мере на 90%, на 95%, на 96%, на 97%, на 98%, на 99% или на 100% идентичны аминокислотной последовательности по всей длине последовательности, или на участке последовательности (например, на непрерывном участке длиной 50, 100, 150 или 200 аминокислот) по сравнению с белком Cas9, встречающимся в природе. В аспектах данного изобретения белок Cas9 по существу идентичен белку, идентифицированному по регистрационному номеру UniProt Q99ZW2, либо варианту или гомологу, по существу идентичному белку с данным регистрационным номером. В аспектах данного изобретения последовательность белка Cas9 по меньшей мере на 75% идентична аминокислотной последовательностью белка, идентифицированного по регистрационному номеру UniProt Q99ZW2. В аспектах данного изобретения последовательность белка Cas9 по меньшей мере на 80% идентична аминокислотной последовательностью белка, идентифицированного по регистрационному номеру UniProt Q99ZW2. В аспектах данного изобретения последовательность белка Cas9 по меньшей мере на 85% идентична аминокислотной последовательностью белка, идентифицированного по регистрационному номеру UniProt Q99ZW2. В аспектах данного изобретения последовательность белка Cas9 по меньшей мере на 90% идентична аминокислотной последовательностью белка, идентифицированного по регистрационному номеру UniProt Q99ZW2. В аспектах данного изобретения последовательность белка Cas9 по меньшей мере на 95% идентична аминокислотной последовательностью белка, идентифицированного по регистрационному номеру UniProt Q99ZW2.
[0096] В вариантах реализации данного изобретения фермент нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК представляет собой «ddCpf1» или «ddCas12a». Термины «Cpf1 с мертвой ДНКазой» или «ddCpf1» относятся к Cpf1 из Acidaminococcus sp. (AsCpf1), содержащему мутацию, которая приводит к инактивации ДНКазной активности в Cpf1. В аспектах данного изобретения ddCpf1 включает в себя мутацию E993A в домене RuvC AsCpf1. В аспектах данного изобретения ddCpf1 по существу не обладает детектируемой эндонуклеазной (например, эндодезоксирибонуклеазной) активностью. В аспектах данного изобретения ddCpf1 включает в себя аминокислотную последовательность с SEQ ID NO: 34. В аспектах данного изобретения ddCpf1 имеет аминокислотную последовательность с SEQ ID NO: 34. В аспектах данного изобретения ddCpf1 имеет аминокислотную последовательность, которая по меньшей мере на 50%, на 55%, на 60%, на 65%, на 70%, на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98%, на 99% или на 100% идентична последовательности с SEQ ID NO: 34. В аспектах данного изобретения ddCpf1 имеет аминокислотную последовательность, которая по меньшей мере на 75% идентична последовательности с SEQ ID NO: 34. В аспектах данного изобретения ddCpf1 имеет аминокислотную последовательность, которая по меньшей мере на 80% идентична последовательности с SEQ ID NO: 34. В аспектах данного изобретения ddCpf1 имеет аминокислотную последовательность, которая по меньшей мере на 85% идентична последовательности с SEQ ID NO: 34. В аспектах данного изобретения ddCpf1 имеет аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 34. В аспектах данного изобретения ddCpf1 имеет аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 34.
[0097] В вариантах реализации данного изобретения фермент нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК представляет собой dLbCpf1. Термин «dLbCpf1» относится к мутированному Cpf1 из бактерии Lachnospiraceae ND2006 (LbCpf1), у которого отсутствует ДНКазная активность. В аспектах данного изобретения dLbCpf1 включает в себя мутацию D832A. В аспектах данного изобретения dLbCpf1 по существу не обладает детектируемой эндонуклеазной (например, эндодезоксирибонуклеазной) активностью. В аспектах данного изобретения dLbCpf1 включает в себя аминокислотную последовательность с SEQ ID NO: 35. В аспектах данного изобретения dLbCpf1 имеет аминокислотную последовательность с SEQ ID NO: 35. В аспектах данного изобретения dLbCpf1 имеет аминокислотную последовательность, которая по меньшей мере на 50%, на 55%, на 60%, на 65%, на 70%, на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98%, на 99% или на 100% идентична последовательности с SEQ ID NO: 35. В аспектах данного изобретения dLbCpf1 имеет аминокислотную последовательность, которая по меньшей мере на 75% идентична последовательности с SEQ ID NO: 35. В аспектах данного изобретения dLbCpf1 имеет аминокислотную последовательность, которая по меньшей мере на 80% идентична последовательности с SEQ ID NO: 35. В аспектах данного изобретения dLbCpf1 имеет аминокислотную последовательность, которая по меньшей мере на 85% идентична последовательности с SEQ ID NO: 35. В аспектах данного изобретения dLbCpf1 имеет аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 35. В аспектах данного изобретения dLbCpf1 имеет аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 35.
[0098] В вариантах реализации данного изобретения фермент нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК представляет собой dFnCpf1. Термин «dFnCpf1» относится к мутированному Cpf1 из Francisella novicida U112 (FnCpf1), у которого отсутствует ДНКазная активность. В аспектах данного изобретения dFnCpf1 включает в себя мутацию D917A. В аспектах данного изобретения dFnCpf1 по существу не обладает детектируемой эндонуклеазной (например, эндодезоксирибонуклеазной) активностью. В аспектах данного изобретения dFnCpf1 включает в себя аминокислотную последовательность с SEQ ID NO: 36. В аспектах данного изобретения dFnCpf1 имеет аминокислотную последовательность с SEQ ID NO: 36. В аспектах данного изобретения dFnCpf1 имеет аминокислотную последовательность, которая по меньшей мере на 50%, на 55%, на 60%, на 65%, на 70%, на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98%, на 99% или на 100% идентична последовательности с SEQ ID NO: 36. В аспектах данного изобретения dFnCpf1 имеет аминокислотную последовательность, которая по меньшей мере на 75% идентична последовательности с SEQ ID NO: 36. В аспектах данного изобретения dFnCpf1 имеет аминокислотную последовательность, которая по меньшей мере на 80% идентична последовательности с SEQ ID NO: 36. В аспектах данного изобретения dFnCpf1 имеет аминокислотную последовательность, которая по меньшей мере на 85% идентична последовательности с SEQ ID NO: 36. В аспектах данного изобретения dFnCpf1 имеет аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 36. В аспектах данного изобретения dFnCpf1 имеет аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 36.
[0099] Термин «Cpf1», или «белок Cpf1», употребляемый в контексте данного документа, включает в себя любую из рекомбинантных или встречающихся в природе форм эндонуклеазы Cpf1 (CRISPR из Prevotella и Francisella 1), или их вариантов или гомологов, которые сохраняют эндонуклеазную активность фермента Cpf1 (например, в пределах по меньшей мере 50%, 80%, 90%, 95%, 96%, 97%, 98%, 99% или 100% активности по сравнению с Cpf1). В аспектах данного изобретения варианты или гомологи по меньшей мере на 90%, на 95%, на 96%, на 97%, на 98%, на 99% или на 100% идентичны аминокислотной последовательности по всей длине последовательности, или на участке последовательности (например, на непрерывном участке длиной 50, 100, 150 или 200 аминокислот) по сравнению с белком Cpf1, встречающимся в природе. В аспектах данного изобретения белок Cpf1 по существу идентичен белку, идентифицированному по регистрационному номеру UniProt U2UMQ6, либо варианту или гомологу, по существу идентичному белку с данным регистрационным номером. В аспектах данного изобретения белок Cpf1 идентичен белку, идентифицированному по регистрационному номеру UniProt U2UMQ6. В аспектах данного изобретения последовательность белка Cpf1 по меньшей мере на 75% идентична аминокислотной последовательности белка, идентифицированного по регистрационному номеру UniProt U2UMQ6. В аспектах данного изобретения последовательность белка Cpf1 по меньшей мере на 80% идентична аминокислотной последовательности белка, идентифицированного по регистрационному номеру UniProt U2UMQ6. В аспектах данного изобретения белок Cpf1 идентичен белку, идентифицированному по регистрационному номеру UniProt U2UMQ6. В аспектах данного изобретения последовательность белка Cpf1 по меньшей мере на 85% идентична аминокислотной последовательности белка, идентифицированного по регистрационному номеру UniProt U2UMQ6. В аспектах данного изобретения белок Cpf1 идентичен белку, идентифицированному по регистрационному номеру UniProt U2UMQ6. В аспектах данного изобретения последовательность белка Cpf1 по меньшей мере на 90% идентична аминокислотной последовательности белка, идентифицированного по регистрационному номеру UniProt U2UMQ6. В аспектах данного изобретения белок Cpf1 идентичен белку, идентифицированному по регистрационному номеру UniProt U2UMQ6. В аспектах данного изобретения последовательность белка Cpf1 по меньшей мере на 95% идентична аминокислотной последовательности белка, идентифицированного по регистрационному номеру UniProt U2UMQ6.
[0100] В вариантах реализации данного изобретения фермент нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК представляет собой нуклеазо-дефицитный вариант Cas9. Термин «нуклеазо-дефицитный вариант Cas9» относится к белку Cas9, имеющему одну или несколько мутаций, которые увеличивают его специфичность связывания с ППМ (англ. «PAM») по сравнению с Cas9 дикого типа, и, кроме того, имеющему мутации, которые делают белок неспособным к эндонуклеазной активности или имеющим серьезно нарушенную эндонуклеазную активность. Не ограничиваясь теорией, считается, что целевая последовательность должна быть связана с ППМ (примыкающий к протоспейсеру мотив); то есть представляет собой короткую последовательность, которая распознается комплексом CRISPR. Требования к точной последовательности и длине для ППМ различаются в зависимости от используемого фермента CRISPR, но ППМ обычно представляют собой последовательности из 2-5 пар оснований, смежные с протоспейсером (то есть с целевой последовательностью). Специфичность связывания нуклеазо-дефицитных вариантов Cas9 с ППМ может быть определена любым методом, известным в данной области техники. Описание и применение известных вариантов Cas9 можно найти, например, в Shmakov et al., Diversity and evolution of class 2 CRISPR-Cas systems. Nat. Rev. Microbiol. 15, 2017, и в Cebrian-Serrano et al, CRISPR-Cas orthologues and variants: optimizing the repertoire, specificity and delivery of genome engineering tools. Mamm. Genome 7-8, 2017, которые во всей своей полноте и для всех целей включены в данный документ посредством ссылки. Иллюстративные варианты Cas9 перечислены в Таблице 4 ниже.
[0101] Таблица 4
(Q695A, Q926A, N497A, R661A)
[0102] В вариантах реализации данного изобретения фермент нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК представляет собой нуклеазо-дефицитную эндонуклеазу CRISPR класса II. Термин «нуклеазо-дефицитная эндонуклеаза CRISPR класса II», употребляемый в контексте данного документа, относится к любой эндонуклеазе CRISPR класса II, имеющей мутации, приводящие к снижению, нарушению или полной потере эндонуклеазной активности.
[0103] В вариантах реализации данного изобретения домен метилтрансферазы ДНК представляет собой домен Dnmt3A-3L. Термин «домен Dnmt3A-3L», употребляемый контексте данного документа, относится к белку, включающему в себя как Dnmt3A, так и Dnmt3L. В аспектах данного изобретения Dnmt3A и Dnmt3L ковалентно связаны. В некоторых аспектах данного изобретения Dnmt3A ковалентно связан с Dnmt3L через пептидный линкер. В аспектах данного изобретения пептидный линкер включает в себя последовательность, изложенную в SEQ ID NO: 27. В аспектах данного изобретения пептидный линкер представляет собой последовательность, изложенную в SEQ ID NO: 27. В аспектах данного изобретения пептидный линкер имеет аминокислотную последовательность, которая по меньшей мере на 50%, на 55%, на 60%, на 65%, на 70%, на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98%, на 99% или на 100% идентична последовательности с SEQ ID NO: 27. В аспектах данного изобретения пептидный линкер имеет аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 27. В аспектах данного изобретения пептидный линкер имеет аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 27. В аспектах данного изобретения домен Dnmt3A-3L включает в себя последовательность, изложенную в SEQ ID NO: 33. В аспектах данного изобретения домен Dnmt3A-3L представляет собой последовательность, изложенную в SEQ ID NO: 33. В аспектах данного изобретения домен Dnmt3A-3L имеет аминокислотную последовательность, которая по меньшей мере на 50%, на 55%, на 60%, на 65%, на 70%, на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98%, на 99% или на 100% идентична последовательности с SEQ ID NO: 33. В аспектах данного изобретения домен Dnmt3A-3L имеет аминокислотную последовательность, которая по меньшей мере на 75% идентична последовательности с SEQ ID NO: 33. В аспектах данного изобретения домен Dnmt3A-3L имеет аминокислотную последовательность, которая по меньшей мере на 80% идентична последовательности с SEQ ID NO: 33. В аспектах данного изобретения домен Dnmt3A-3L имеет аминокислотную последовательность, которая по меньшей мере на 85% идентична последовательности с SEQ ID NO: 33. В аспектах данного изобретения домен Dnmt3A-3L имеет аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 33. В аспектах данного изобретения домен Dnmt3A-3L имеет аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 33.
[0104] В вариантах реализации данного изобретения пептидный линкер представляет собой линкер XTEN. В аспектах данного изобретения линкер XTEN включает в себя от около 16 до около 80 аминокислотных остатков. В аспектах данного изобретения линкер XTEN включает в себя от около 17 до около 80 аминокислотных остатков. В аспектах данного изобретения линкер XTEN включает в себя от около 18 до около 80 аминокислотных остатков. В аспектах данного изобретения линкер XTEN включает в себя от около 19 до около 80 аминокислотных остатков. В аспектах данного изобретения линкер XTEN включает в себя от около 20 до около 80 аминокислотных остатков. В аспектах данного изобретения линкер XTEN включает в себя от около 30 до около 80 аминокислотных остатков. В аспектах данного изобретения линкер XTEN включает в себя от около 40 до около 80 аминокислотных остатков. В аспектах данного изобретения линкер XTEN включает в себя от около 50 до около 80 аминокислотных остатков. В аспектах данного изобретения линкер XTEN включает в себя от около 60 до около 80 аминокислотных остатков. В аспектах данного изобретения линкер XTEN включает в себя от около 70 до около 80 аминокислотных остатков. В аспектах данного изобретения линкер XTEN включает в себя от около 16 до около 70 аминокислотных остатков. В аспектах данного изобретения линкер XTEN включает в себя от около 16 до около 60 аминокислотных остатков. В аспектах данного изобретения линкер XTEN включает в себя от около 16 до около 50 аминокислотных остатков. В аспектах данного изобретения линкер XTEN включает в себя от около 16 до около 40 аминокислотных остатков. В аспектах данного изобретения линкер XTEN включает в себя от около 16 до около 35 аминокислотных остатков. В аспектах данного изобретения линкер XTEN включает в себя от около 16 до около 30 аминокислотных остатков. В аспектах данного изобретения линкер XTEN включает в себя от около 16 до около 25 аминокислотных остатков. В аспектах данного изобретения линкер XTEN включает в себя от около 16 до около 20 аминокислотных остатков. В аспектах данного изобретения линкер XTEN включает в себя около 16 аминокислотных остатков. В аспектах данного изобретения линкер XTEN включает в себя около 17 аминокислотных остатков. В аспектах данного изобретения линкер XTEN включает в себя около 18 аминокислотных остатков. В аспектах данного изобретения линкер XTEN включает в себя около 19 аминокислотных остатков. В аспектах данного изобретения линкер XTEN включает в себя около 20 аминокислотных остатков.
[0105] В вариантах реализации данного изобретения линкер XTEN включает в себя последовательность, изложенную в SEQ ID NO: 31. В аспектах данного изобретения линкер XTEN представляет собой последовательность, изложенную в SEQ ID NO: 31. В аспектах данного изобретения линкер XTEN включает в себя последовательность, изложенную SEQ ID NO: 32. В аспектах данного изобретения линкер XTEN представляет собой последовательность, изложенную в SEQ ID NO: 32. В аспектах данного изобретения линкер XTEN имеет аминокислотную последовательность, которая по меньшей мере на 50%, на 55%, на 60%, на 65%, на 70%, на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98%, на 99% или на 100% идентична последовательности с SEQ ID NO: 31. В аспектах данного изобретения линкер XTEN имеет аминокислотную последовательность, которая по меньшей мере на 85% идентична последовательности с SEQ ID NO: 31. В аспектах данного изобретения линкер XTEN имеет аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 31. В аспектах данного изобретения линкер XTEN имеет аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 31. В аспектах данного изобретения линкер XTEN имеет аминокислотную последовательность, которая по меньшей мере на 50%, на 55%, на 60%, на 65%, на 70%, на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98%, на 99% или на 100% идентична последовательности с SEQ ID NO: 32. В аспектах данного изобретения линкер XTEN имеет аминокислотную последовательность, которая по меньшей мере на 75% идентична последовательности с SEQ ID NO: 32. В аспектах данного изобретения линкер XTEN имеет аминокислотную последовательность, которая по меньшей мере на 80% идентична последовательности с SEQ ID NO: 32. В аспектах данного изобретения линкер XTEN имеет аминокислотную последовательность, которая по меньшей мере на 85% идентична последовательности с SEQ ID NO: 32. В аспектах данного изобретения линкер XTEN имеет аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 32. В аспектах данного изобретения линкер XTEN имеет аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 32.
[0106] Слитый белок может включать в себя аминокислотные последовательности, пригодные для нацеливания слитого белка на определенные области клетки (например, цитоплазму, ядро). Таким образом, в аспектах данного изобретения слитый белок дополнительно включает в себя пептид сигнала ядерной локализации (СЯЛ). В аспектах данного изобретения СЯЛ включает в себя последовательность, изложенную в SEQ ID NO: 25. В аспектах данного изобретения СЯЛ представляет собой последовательность, изложенную в SEQ ID NO: 25. В аспектах данного изобретения СЯЛ имеет аминокислотную последовательность, которая по меньшей мере на 50%, на 55%, на 60%, на 65%, на 70%, на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98%, на 99% или на 100% идентична последовательности с SEQ ID NO: 25. В аспектах данного изобретения СЯЛ имеет аминокислотную последовательность, которая по меньшей мере на 75% идентична последовательности с SEQ ID NO: 25. В аспектах данного изобретения СЯЛ имеет аминокислотную последовательность, которая по меньшей мере на 80% идентична последовательности с SEQ ID NO: 25. В аспектах данного изобретения СЯЛ имеет аминокислотную последовательность, которая по меньшей мере на 85% идентична последовательности с SEQ ID NO: 25. В аспектах данного изобретения СЯЛ имеет аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 25. В аспектах данного изобретения СЯЛ имеет аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 25.
[0107] В вариантах реализации данного изобретения слитый белок включает в себя в направлении от N-конца до C-конца домен KRAB, фермент нуклеазо-дефицитную РНК-направляемую эндонуклеазу ДНК и домен метилтрансферазы ДНК.
[0108] В вариантах реализации данного изобретения фермент нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК представляет собой dCas9, а домен метилтрансферазы ДНК представляет собой домен Dnmt3A-3L.
[0109] В вариантах реализации данного изобретения dCas9 ковалентно связан с доменом KRAB через пептидный линкер, и при этом dCas9 ковалентно связан с доменом Dnmt3A-3L через пептидный линкер.
[0110] В вариантах реализации данного изобретения пептидный линкер представляет собой линкер XTEN. В аспектах данного изобретения линкер XTEN включает в себя последовательность, изложенную в SEQ ID NO: 31. В аспектах данного изобретения линкер XTEN представляет собой последовательность, изложенную в SEQ ID NO: 31. В аспектах данного изобретения линкер XTEN имеет аминокислотную последовательность, которая по меньшей мере на 75% идентична последовательности с SEQ ID NO: 31. В аспектах данного изобретения линкер XTEN имеет аминокислотную последовательность, которая по меньшей мере на 80% идентична последовательности с SEQ ID NO: 31. В аспектах данного изобретения линкер XTEN имеет аминокислотную последовательность, которая по меньшей мере на 85% идентична последовательности с SEQ ID NO: 31. В аспектах данного изобретения линкер XTEN имеет аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 31. В аспектах данного изобретения линкер XTEN имеет аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 31. В аспектах данного изобретения линкер XTEN включает в себя последовательность, изложенную SEQ ID NO: 32. В аспектах данного изобретения линкер XTEN представляет собой последовательность, изложенную в SEQ ID NO: 32. В аспектах данного изобретения линкер XTEN имеет аминокислотную последовательность, которая по меньшей мере на 75% идентична последовательности с SEQ ID NO: 32. В аспектах данного изобретения линкер XTEN имеет аминокислотную последовательность, которая по меньшей мере на 80% идентична последовательности с SEQ ID NO: 32. В аспектах данного изобретения линкер XTEN имеет аминокислотную последовательность, которая по меньшей мере на 85% идентична последовательности с SEQ ID NO: 32. В аспектах данного изобретения линкер XTEN имеет аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 32. В аспектах данного изобретения линкер XTEN имеет аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 32.
[0111] В вариантах реализации данного изобретения слитый белок включает в себя аминокислотную последовательность с SEQ ID NO: 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14 или 15. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность с SEQ ID NO: 1. В аспектах данного изобретения слитый белок представляет собой аминокислотную последовательность с SEQ ID NO: 1. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность с SEQ ID NO: 2. В аспектах данного изобретения слитый белок представляет собой аминокислотную последовательность с SEQ ID NO: 2. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность с SEQ ID NO: 3. В аспектах данного изобретения слитый белок представляет собой аминокислотную последовательность с SEQ ID NO: 3. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность с SEQ ID NO: 4. В аспектах данного изобретения слитый белок представляет собой аминокислотную последовательность с SEQ ID NO: 4. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность с SEQ ID NO: 5. В аспектах данного изобретения слитый белок представляет собой аминокислотную последовательность с SEQ ID NO: 5. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность с SEQ ID NO: 6. В аспектах данного изобретения слитый белок представляет собой аминокислотную последовательность с SEQ ID NO: 6. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность с SEQ ID NO: 7. В аспектах данного изобретения слитый белок представляет собой аминокислотную последовательность с SEQ ID NO: 7. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность с SEQ ID NO: 8. В аспектах данного изобретения слитый белок представляет собой аминокислотную последовательность с SEQ ID NO: 8. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность с SEQ ID NO: 9. В аспектах данного изобретения слитый белок представляет собой аминокислотную последовательность с SEQ ID NO: 9. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность с SEQ ID NO: 10. В аспектах данного изобретения слитый белок представляет собой аминокислотную последовательность с SEQ ID NO: 10. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность с SEQ ID NO: 11. В аспектах данного изобретения слитый белок представляет собой аминокислотную последовательность с SEQ ID NO: 11. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность с SEQ ID NO: 12. В аспектах данного изобретения слитый белок представляет собой аминокислотную последовательность с SEQ ID NO: 12. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность с SEQ ID NO: 13. В аспектах данного изобретения слитый белок представляет собой аминокислотную последовательность с SEQ ID NO: 13. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность с SEQ ID NO: 14. В аспектах данного изобретения слитый белок представляет собой аминокислотную последовательность с SEQ ID NO: 14. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность с SEQ ID NO: 15. В аспектах данного изобретения слитый белок представляет собой аминокислотную последовательность с SEQ ID NO: 15.
[0112] В вариантах реализации данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 75%, на 80%, на 81%, на 82%, на 83%, на 84%, на 85%, на 86%, на 87%, на 88%, на 89%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14 или 15. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 75%, на 80%, на 81%, на 82%, на 83%, на 84%, на 85%, на 86%, на 87%, на 88%, на 89%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 1. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 75%, на 80%, на 81%, на 82%, на 83%, на 84%, на 85%, на 86%, на 87%, на 88%, на 89%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 2. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 75%, на 80%, на 81%, на 82%, на 83%, на 84%, на 85%, на 86%, на 87%, на 88%, на 89%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 3. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 75%, на 80%, на 81%, на 82%, на 83%, на 84%, на 85%, на 86%, на 87%, на 88%, на 89%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 4. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 75%, на 80%, на 81%, на 82%, на 83%, на 84%, на 85%, на 86%, на 87%, на 88%, на 89%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 5. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 75%, на 80%, на 81%, на 82%, на 83%, на 84%, на 85%, на 86%, на 87%, на 88%, на 89%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 6. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 75%, на 80%, на 81%, на 82%, на 83%, на 84%, на 85%, на 86%, на 87%, на 88%, на 89%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 7. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 75%, на 80%, на 81%, на 82%, на 83%, на 84%, на 85%, на 86%, на 87%, на 88%, на 89%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 8. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 75%, на 80%, на 81%, на 82%, на 83%, на 84%, на 85%, на 86%, на 87%, на 88%, на 89%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 9. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 75%, на 80%, на 81%, на 82%, на 83%, на 84%, на 85%, на 86%, на 87%, на 88%, на 89%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 10. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 75%, на 80%, на 81%, на 82%, на 83%, на 84%, на 85%, на 86%, на 87%, на 88%, на 89%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 11. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 75%, на 80%, на 81%, на 82%, на 83%, на 84%, на 85%, на 86%, на 87%, на 88%, на 89%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 12. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 75%, на 80%, на 81%, на 82%, на 83%, на 84%, на 85%, на 86%, на 87%, на 88%, на 89%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 13. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 75%, на 80%, на 81%, на 82%, на 83%, на 84%, на 85%, на 86%, на 87%, на 88%, на 89%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 14. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 75%, на 80%, на 81%, на 82%, на 83%, на 84%, на 85%, на 86%, на 87%, на 88%, на 89%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 15.
[0113] В вариантах реализации данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 75% идентична последовательности с SEQ ID NO: 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14 или 15. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 75% идентична последовательности с SEQ ID NO: 1. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 75% идентична последовательности с SEQ ID NO: 2. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 75% идентична последовательности с SEQ ID NO: 3. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 75% идентична последовательности с SEQ ID NO: 4. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 75% идентична последовательности с SEQ ID NO: 5. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 75% идентична последовательности с SEQ ID NO: 6. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 75% идентична последовательности с SEQ ID NO: 7. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 75% идентична последовательности с SEQ ID NO: 8. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 75% идентична последовательности с SEQ ID NO: 9. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 75% идентична последовательности с SEQ ID NO: 10. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 75% идентична последовательности с SEQ ID NO: 11. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 75% идентична последовательности с SEQ ID NO: 12. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 75% идентична последовательности с SEQ ID NO: 13. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 75% идентична последовательности с SEQ ID NO: 14. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 75% идентична последовательности с SEQ ID NO: 15.
[0114] В вариантах реализации данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 80% идентична последовательности с SEQ ID NO: 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14 или 15. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 80% идентична последовательности с SEQ ID NO: 1. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 80% идентична последовательности с SEQ ID NO: 2. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 80% идентична последовательности с SEQ ID NO: 3. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 80% идентична последовательности с SEQ ID NO: 4. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 80% идентична последовательности с SEQ ID NO: 5. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 80% идентична последовательности с SEQ ID NO: 6. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 80% идентична последовательности с SEQ ID NO: 7. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 80% идентична последовательности с SEQ ID NO: 8. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 80% идентична последовательности с SEQ ID NO: 9. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 80% идентична последовательности с SEQ ID NO: 10. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 80% идентична последовательности с SEQ ID NO: 11. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 80% идентична последовательности с SEQ ID NO: 12. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 80% идентична последовательности с SEQ ID NO: 13. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 80% идентична последовательности с SEQ ID NO: 14. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 80% идентична последовательности с SEQ ID NO: 15.
[0115] В вариантах реализации данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 85% идентична последовательности с SEQ ID NO: 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14 или 15. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 85% идентична последовательности с SEQ ID NO: 1. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 85% идентична последовательности с SEQ ID NO: 2. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 85% идентична последовательности с SEQ ID NO: 3. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 85% идентична последовательности с SEQ ID NO: 4. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 85% идентична последовательности с SEQ ID NO: 5. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 85% идентична последовательности с SEQ ID NO: 6. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 85% идентична последовательности с SEQ ID NO: 7. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 85% идентична последовательности с SEQ ID NO: 8. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 85% идентична последовательности с SEQ ID NO: 9. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 85% идентична последовательности с SEQ ID NO: 10. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 85% идентична последовательности с SEQ ID NO: 11. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 85% идентична последовательности с SEQ ID NO: 12. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 85% идентична последовательности с SEQ ID NO: 13. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 85% идентична последовательности с SEQ ID NO: 14. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 85% идентична последовательности с SEQ ID NO: 15.
[0116] В вариантах реализации данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14 или 15. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 1. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 2. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 3. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 4. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 5. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 6. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 7. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 8. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 9. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 10. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 11. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 12. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 13. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 14. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 90% идентична последовательности с SEQ ID NO: 15.
[0117] В вариантах реализации данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14 или 15. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 1. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 2. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 3. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 4. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 5. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 6. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 7. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 8. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 9. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 10. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 11. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 12. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 13. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 14. В аспектах данного изобретения слитый белок включает в себя аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности с SEQ ID NO: 15.
[0118] Комплексы
[0119] Для того, чтобы слитый белок мог выполнять редактирование эпигенома, слитый белок взаимодействует с (например, нековалентно связан с) полинуклеотидом (например, егРНК), который комплементарен целевой полинуклеотидной последовательности (например, целевой последовательности ДНК, которая должна быть отредактирована) и, кроме того, включает в себя последовательность (т.е. последовательность связывания), с которой может связываться фермент нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК, как описано в данном документе. Путем образования такого комплекса, гибридный белок соответствующим образом позиционируется для выполнения редактирования эпигенома. Термин «комплекс» относится к композиции, которая включает в себя два или более компонента, при этом компоненты связываются вместе и образуют функциональную единицу. В аспектах данного изобретения описанный в данном документе комплекс включает в себя слитый белок, описанный в данном документе, и полинуклеотид, описанный в данном документе. Таким образом, в одном аспекте данного изобретения представлен слитый белок, как описано в данном документе, включительно с его вариантами реализации и аспектами, и полинуклеотид, включающий в себя: (1) нацеливающую на ДНК последовательность, которая комплементарна целевой полинуклеотидной последовательности; и (2) связывающую последовательность для фермента нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК, при этом фермент нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК связан с полинуклеотидом через связывающую последовательность (например, аминокислотную последовательность, способную связываться с нацеливающей последовательностью ДНК).
[0120] Нацеливающая на ДНК последовательность относится к полинуклеотиду, который включает в себя нуклеотидную последовательность, комплементарную целевой полинуклеотидной последовательности (ДНК или РНК). В аспектах данного изобретения нацеливающая на ДНК последовательность может быть единой молекулой РНК (одиночным полинуклеотидом РНК), которая может включать в себя «единую гидовую РНК» или «егРНК». В аспектах данного изобретения нацеливающая на ДНК последовательность включает в себя две молекулы РНК (например, соединенные вместе посредством гибридизации по месту связывающей последовательности (например, по месту dCas9-связывающей последовательности). В аспектах данного изобретения нацеливающая на ДНК последовательность (например, егРНК) по меньшей мере на 50%, на 55%, на 60%, на 65%, на 70%, на 75%, на 80%, на 85%, на 90%, на 95%, на 96%, на 97%, на 98% или на 99% комплементарна целевой полинуклеотидной последовательности. В аспектах данного изобретения нацеливающая на ДНК последовательность (например, егРНК) по меньшей мере на 50%, на 55%, на 60%, на 65%, на 70%, на 75%, на 80%, на 85%, на 90%, на 95%, на 96%, на 97%, на 98% или на 99% комплементарна последовательности клеточного гена. В аспектах данного изобретения нацеливающая на ДНК последовательность (например, егРНК) связывает последовательность клеточного гена. В аспектах данного изобретения нацеливающая на ДНК последовательность (например, егРНК) по крайней мере на 75% комплементарна последовательности клеточного гена. В аспектах данного изобретения нацеливающая на ДНК последовательность (например, егРНК) по крайней мере на 80% комплементарна последовательности клеточного гена. В аспектах данного изобретения нацеливающая на ДНК последовательность (например, егРНК) связывает последовательность клеточного гена. В аспектах данного изобретения нацеливающая на ДНК последовательность (например, егРНК) по крайней мере на 85% комплементарна последовательности клеточного гена. В аспектах данного изобретения нацеливающая на ДНК последовательность (например, егРНК) связывает последовательность клеточного гена. В аспектах данного изобретения нацеливающая на ДНК последовательность (например, егРНК) по крайней мере на 90% комплементарна последовательности клеточного гена. В аспектах данного изобретения нацеливающая на ДНК последовательность (например, егРНК) связывает последовательность клеточного гена. В аспектах данного изобретения нацеливающая на ДНК последовательность (например, егРНК) по крайней мере на 95% комплементарна последовательности клеточного гена. В аспектах данного изобретения нацеливающая на ДНК последовательность (например, егРНК) связывает последовательность клеточного гена.
[0121] «Целевая полинуклеотидная последовательность», как предусмотрено в данном документе, представляет собой последовательность нуклеиновой кислоты, которая присутствует в клетке или экспрессируется ею, для которой сконструирована комплементарная направляющая последовательность (или нацеливающая на ДНК последовательность), при этом гибридизация между целевой последовательностью и направляющей последовательностью (или нацеливающей на ДНК последовательностью) способствует образованию комплекса CRISPR. Полная комплементарность не обязательно требуется при условии, что комплементарность достаточна, чтобы вызвать гибридизацию и способствовать образованию комплекса CRISPR. В аспектах данного изобретения целевая полинуклеотидная последовательность представляет собой экзогенную последовательность нуклеиновой кислоты. В аспектах данного изобретения целевая полинуклеотидная последовательность представляет собой эндогенную последовательность нуклеиновой кислоты.
[0122] Целевая полинуклеотидная последовательность может представлять собой любую область полинуклеотида (например, последовательность ДНК), подходящую для редактирования эпигенома. В аспектах данного изобретения целевая полинуклеотидная последовательность представляет собой часть гена. В аспектах данного изобретения целевая полинуклеотидная последовательность представляет собой часть транскрипционной регуляторной последовательности. В аспектах данного изобретения целевая полинуклеотидная последовательность представляет собой часть промотора, энхансера или сайленсера. В аспектах данного изобретения целевая полинуклеотидная последовательность представляет собой часть промотора. В аспектах данного изобретения целевая полинуклеотидная последовательность представляет собой часть энхансера. В аспектах данного изобретения целевая полинуклеотидная последовательность представляет собой часть сайленсера.
[0123] В вариантах реализации данного изобретения целевая полинуклеотидная последовательность представляет собой последовательность гипометилированной нуклеиновой кислоты. Термин «последовательность гипометилированной нуклеиновой кислоты» употребляется в контексте данного документа в соответствии со стандартным значением в данной области техники и относится к потере или отсутствию метильных групп в 5-метилцитозиновом нуклеотиде (например, в CpG). Потеря или отсутствие метильных групп может быть относительно стандартного контроля. Гипометилирование может происходить, например, в стареющих клетках или при онкологическом заболевании (например, на ранних стадиях неоплазии) по сравнению с более молодой клеткой или нераковой клеткой, соответственно. Таким образом, комплекс может быть полезен для восстановления нормальных (например, незрелых или здоровых) уровней метилирования.
[0124] В вариантах реализации данного изобретения целевая полинуклеотидная последовательность находится в пределах около 3000 пар оснований (п.о.), фланкирующих сайт начала транскрипции. В аспектах данного изобретения целевая полинуклеотидная последовательность находится в пределах около 3000, около 2900, около 2800, около 2700, около 2600, около 2500, около 2400, около 2300, около 2200, около 2100, около 2000, около 1900, около 1800, около 1700, около 1600, около 1500, около 1400, около 1300, около 1200, около 1100, около 1000, около 900, около 800, около 700, около 600, около 500, около 400, около 300, около 200 или около 100 пар оснований (п.о.), фланкирующих сайт начала транскрипции.
[0125] В вариантах реализации данного изобретения целевая полинуклеотидная последовательность находится рядом с промоторной последовательностью, рядом с ней или внутри нее. В аспектах данного изобретения целевая полинуклеотидная последовательность находится внутри островка CpG. В аспектах данного изобретения известно, что целевая полинуклеотидная последовательность связана с заболеванием или патологическим состоянием, характеризующимся гипометилированием ДНК.
[0126] В вариантах реализации данного изобретения иллюстративные целевые полинуклеотидные последовательности включают в себя последовательности, описанные в Таблицах 1 и 2. В аспектах данного изобретения целевая полинуклеотидная последовательность включает в себя последовательность с SEQ ID NO: 37, 39, 41, 43, 45, 47, 49, 51, 53, 55, 57, 59, 61, 63, 65, 67, 69, 71, 73, 75, 77, 79, 81, 83, 85, 87, 89, 91, 93 или 95. В аспектах данного изобретения целевая полинуклеотидная последовательность включает в себя аминокислотную последовательность, которая по меньшей мере на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 37, 39, 41, 43, 45, 47, 49, 51, 53, 55, 57, 59, 61, 63, 65, 67, 69, 71, 73, 75, 77, 79, 81, 83, 85, 87, 89, 91, 93 или 95. В аспектах данного изобретения целевая полинуклеотидная последовательность представляет собой последовательность с SEQ ID NO: 37. В аспектах данного изобретения целевая полинуклеотидная последовательность представляет собой последовательность с SEQ ID NO: 39. В аспектах данного изобретения целевая полинуклеотидная последовательность представляет собой последовательность с SEQ ID NO: 41. В аспектах данного изобретения целевая полинуклеотидная последовательность представляет собой последовательность с SEQ ID NO: 43. В аспектах данного изобретения целевая полинуклеотидная последовательность представляет собой последовательность с SEQ ID NO: 45. В аспектах данного изобретения целевая полинуклеотидная последовательность представляет собой последовательность с SEQ ID NO: 47. В аспектах данного изобретения целевая полинуклеотидная последовательность представляет собой последовательность с SEQ ID NO: 49. В аспектах данного изобретения целевая полинуклеотидная последовательность представляет собой последовательность с SEQ ID NO: 51. В аспектах данного изобретения целевая полинуклеотидная последовательность представляет собой последовательность с SEQ ID NO: 53. В аспектах данного изобретения целевая полинуклеотидная последовательность представляет собой последовательность с SEQ ID NO: 55. В аспектах данного изобретения целевая полинуклеотидная последовательность представляет собой последовательность с SEQ ID NO: 57. В аспектах данного изобретения целевая полинуклеотидная последовательность представляет собой последовательность с SEQ ID NO: 59. В аспектах данного изобретения целевая полинуклеотидная последовательность представляет собой последовательность с SEQ ID NO: 61. В аспектах данного изобретения целевая полинуклеотидная последовательность представляет собой последовательность с SEQ ID NO: 63. В аспектах данного изобретения целевая полинуклеотидная последовательность представляет собой последовательность с SEQ ID NO: 65. В аспектах данного изобретения целевая полинуклеотидная последовательность представляет собой последовательность с SEQ ID NO: 67. В аспектах данного изобретения целевая полинуклеотидная последовательность представляет собой последовательность с SEQ ID NO: 69. В аспектах данного изобретения целевая полинуклеотидная последовательность представляет собой последовательность с SEQ ID NO: 71. В аспектах данного изобретения целевая полинуклеотидная последовательность представляет собой последовательность с SEQ ID NO: 73. В аспектах данного изобретения целевая полинуклеотидная последовательность представляет собой последовательность с SEQ ID NO: 75. В аспектах данного изобретения целевая полинуклеотидная последовательность представляет собой последовательность с SEQ ID NO: 77. В аспектах данного изобретения целевая полинуклеотидная последовательность представляет собой последовательность с SEQ ID NO: 79. В аспектах данного изобретения целевая полинуклеотидная последовательность представляет собой последовательность с SEQ ID NO: 81. В аспектах данного изобретения целевая полинуклеотидная последовательность представляет собой последовательность с SEQ ID NO: 83. В аспектах данного изобретения целевая полинуклеотидная последовательность представляет собой последовательность с SEQ ID NO: 85. В аспектах данного изобретения целевая полинуклеотидная последовательность представляет собой последовательность с SEQ ID NO: 87. В аспектах данного изобретения целевая полинуклеотидная последовательность представляет собой последовательность с SEQ ID NO: 89. В аспектах данного изобретения целевая полинуклеотидная последовательность представляет собой последовательность с SEQ ID NO: 91. В аспектах данного изобретения целевая полинуклеотидная последовательность представляет собой последовательность с SEQ ID NO: 93. В аспектах данного изобретения целевая полинуклеотидная последовательность представляет собой последовательность с SEQ ID NO: 95.
[0127] В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 37. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 39. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 41. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 43. В аспектах данного изобретения целевая полинуклеотидная последовательность представляет собой последовательность с SEQ ID NO: 45. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 47. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 49. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 51. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 53. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 55. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 57. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 59. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 61. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 63. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 65. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 67. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 69. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 71. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 73. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 75. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 77. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 79. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 81. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 83. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 85. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 87. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 89. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 91. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 93. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 75%, на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 95.
[0128] В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 90% идентична последовательности с SEQ ID NO: 37. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 90% идентична последовательности с SEQ ID NO: 39. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 90% идентична последовательности с SEQ ID NO: 41. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 90% идентична последовательности с SEQ ID NO: 43. В аспектах данного изобретения целевая полинуклеотидная последовательность представляет собой последовательность с SEQ ID NO: 45. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 90% идентична последовательности с SEQ ID NO: 47. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 90% идентична последовательности с SEQ ID NO: 49. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 90% идентична последовательности с SEQ ID NO: 51. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 90% идентична последовательности с SEQ ID NO: 53. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 90% идентична последовательности с SEQ ID NO: 55. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 90% идентична последовательности с SEQ ID NO: 57. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 90% идентична последовательности с SEQ ID NO: 59. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 90% идентична последовательности с SEQ ID NO: 61. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 90% идентична последовательности с SEQ ID NO: 63. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 90% идентична последовательности с SEQ ID NO: 65. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 90% идентична последовательности с SEQ ID NO: 67. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 90% идентична последовательности с SEQ ID NO: 69. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 90% идентична последовательности с SEQ ID NO: 71. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 90% идентична последовательности с SEQ ID NO: 73. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 90% идентична последовательности с SEQ ID NO: 75. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 90% идентична последовательности с SEQ ID NO: 77. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 90% идентична последовательности с SEQ ID NO: 79. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 90% идентична последовательности с SEQ ID NO: 81. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 90% идентична последовательности с SEQ ID NO: 83. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 90% идентична последовательности с SEQ ID NO: 85. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 90% идентична последовательности с SEQ ID NO: 87. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 90% идентична последовательности с SEQ ID NO: 89. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 90% идентична последовательности с SEQ ID NO: 91. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 90% идентична последовательности с SEQ ID NO: 93. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 90% идентична последовательности с SEQ ID NO: 95.
[0129] В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 95% идентична последовательности с SEQ ID NO: 37. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 95% идентична последовательности с SEQ ID NO: 39. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 95% идентична последовательности с SEQ ID NO: 41. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 95% идентична последовательности с SEQ ID NO: 43. В аспектах данного изобретения целевая полинуклеотидная последовательность представляет собой последовательность с SEQ ID NO: 45. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 95% идентична последовательности с SEQ ID NO: 47. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 95% идентична последовательности с SEQ ID NO: 49. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 95% идентична последовательности с SEQ ID NO: 51. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 95% идентична последовательности с SEQ ID NO: 53. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 95% идентична последовательности с SEQ ID NO: 55. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 95% идентична последовательности с SEQ ID NO: 57. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 95% идентична последовательности с SEQ ID NO: 59. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 95% идентична последовательности с SEQ ID NO: 61. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 95% идентична последовательности с SEQ ID NO: 63. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 95% идентична последовательности с SEQ ID NO: 65. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 95% идентична последовательности с SEQ ID NO: 67. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 95% идентична последовательности с SEQ ID NO: 69. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 95% идентична последовательности с SEQ ID NO: 71. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 95% идентична последовательности с SEQ ID NO: 73. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 95% идентична последовательности с SEQ ID NO: 75. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 95% идентична последовательности с SEQ ID NO: 77. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 95% идентична последовательности с SEQ ID NO: 79. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 95% идентична последовательности с SEQ ID NO: 81. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 95% идентична последовательности с SEQ ID NO: 83. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 95% идентична последовательности с SEQ ID NO: 85. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 95% идентична последовательности с SEQ ID NO: 87. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 95% идентична последовательности с SEQ ID NO: 89. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 95% идентична последовательности с SEQ ID NO: 91. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 95% идентична последовательности с SEQ ID NO: 93. В аспектах данного изобретения целевая полинуклеотидная последовательность по меньшей мере на 95% идентична последовательности с SEQ ID NO: 95.
[0130] В вариантах реализации данного изобретения комплекс включает в себя dCas9, связанный с полинуклеотидом посредством связывания связывающей последовательности полинуклеотида и формирующий, таким образом, рибонуклеопротеидный комплекс. В аспектах данного изобретения связывающая последовательность образует шпилечную структуру. В аспектах данного изобретения связывающая последовательность составляет в длину 30-100 нуклеотидов, 35-50 нуклеотидов, 37-47 нуклеотидов или 42 нуклеотида.
[0131] В вариантах реализации данного изобретения связывающая последовательность (например, Cas9-связывающая последовательность) взаимодействует или связывается с белком Cas9 (например, с белком dCas9), и вместе они связываются с целевой полинуклеотидной последовательностью, которая распознается нацеливающей на ДНК последовательностью. Связывающая последовательность (например, Cas9-связывающая последовательность) включает в себя два комплементарных участка нуклеотидов, которые гибридизуются друг с другом с образованием дуплекса двухцепочечной РНК (дуплекс дцРНК). Эти два комплементарных участка нуклеотидов могут быть ковалентно связаны посредством промежуточных нуклеотидов, известных как линкеры или линкерные нуклеотиды (например, в случае одномолекулярного полинуклеотида), и гибридизоваться с образованием дуплекса двухцепочечной РНК (дуплекс дцРНК, или «Cas9-связывающая шпилька») связывающей последовательности (например, Cas9-связывающей последовательности), что приводит к формированию структуры стержень - петля. Альтернативно, в некоторых аспектах данного изобретения два комплементарных участка нуклеотидов могут не быть ковалентно связаны, а вместо этого удерживаются вместе посредством гибридизации между комплементарными последовательностями (например, в случае двухмолекулярного полинуклеотида).
[0132] Связывающая последовательность (например, Cas9-связывающая последовательность) может иметь длину от 10 нуклеотидов до 100 нуклеотидов, например, от 10 нуклеотидов (нт) до 20 нт, от 20 нт до 30 нт, от 30 нт до 40 нт, от 40 нт до 50 нт, от 50 нт до 60 нт, от 60 нт до 70 нт, от 70 нт до 80 нт, от 80 нт до 90 нт или от 90 нт до 100 нт. В аспектах данного изобретения связывающая последовательность имеет длину от 15 нуклеотидов (нт) до 80 нт. В аспектах данного изобретения связывающая последовательность имеет длину от 15 нт до 50 нт. В аспектах данного изобретения связывающая последовательность имеет длину от 15 нт до 40 нт. В аспектах данного изобретения связывающая последовательность имеет длину от 15 нт до 30 нт. В аспектах данного изобретения связывающая последовательность имеет длину от 37 нт до 47 нт (например, 42 нт). В аспектах данного изобретения связывающая последовательность имеет длину от 15 нт до 25 нт.
[0133] Дуплекс дцРНК связывающей последовательности (например, Cas9-связывающей последовательности) может иметь длину от 6 пар оснований (п.о.) до 50 п.о. Например, дуплекс дцРНК связывающей последовательности (например, Cas9-связывающей последовательности) может иметь длину от 6 п.о. до 40 п.о., от 6 п.о. до 30 п.о., от 6 п.о. до 25 п.о., от 6 п.о. до 20 п.о., от 6 п.о. до 15 п.о., от 8 п.о. до 40 п.о., от 8 п.о. до 30 п.о., от 8 п.о. до 25 п.о., от 8 п.о. до 20 п.о. или от 8 п.о. до 15 п.о. В аспектах данного изобретения дуплекс дцРНК связывающей последовательности (например, Cas9-связывающей последовательности) имеет длину от 8 п.о. до 10 п.о. В аспектах данного изобретения дуплекс дцРНК связывающей последовательности (например, Cas9-связывающей последовательности) имеет длину от 10 п.о. до 15 п.о. В аспектах данного изобретения дуплекс дцРНК связывающей последовательности (например, Cas9-связывающей последовательности) имеет длину от 15 п.о. до 18 п.о. В аспектах данного изобретения дуплекс дцРНК связывающей последовательности (например, Cas9-связывающей последовательности) имеет длину от 18 п.о. до 20 п.о. В аспектах данного изобретения дуплекс дцРНК связывающей последовательности (например, Cas9-связывающей последовательности) имеет длину от 20 п.о. до 25 п.о. В аспектах данного изобретения дуплекс дцРНК связывающей последовательности (например, Cas9-связывающей последовательности) имеет длину от 25 п.о. до 30 п.о. В аспектах данного изобретения дуплекс дцРНК связывающей последовательности (например, Cas9-связывающей последовательности) имеет длину от 30 п.о. до 35 п.о. В аспектах данного изобретения дуплекс дцРНК связывающей последовательности (например, Cas9-связывающей последовательности) имеет длину от 35 п.о. до 40 п.о. В аспектах данного изобретения дуплекс дцРНК связывающей последовательности (например, Cas9-связывающей последовательности) имеет длину от 40 п.о. до 50 п.о.
[0134] В вариантах реализации данного изобретения иллюстративный полинуклеотид, который образует комплекс со слитым белком, описанным в данном документе, включает в себя те полинуклеотиды, которые описаны в Таблицах 1 и 2 как егРНК. В аспектах данного изобретения полинуклеотид, который образует комплекс со слитым белком, описанным в данном документе, включает в себя последовательность с SEQ ID NO: 38, 40, 42, 44, 46, 48, 50, 52, 54, 56, 58, 60, 62, 64, 66, 68, 70, 72, 74, 76, 78, 80, 82, 84, 86, 88, 90, 92 или 94, или их соответствующие последовательности РНК. В аспектах данного изобретения полинуклеотид, который образует комплекс со слитым белком, описанным в данном документе, включает в себя последовательность, которая по меньшей мере на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98% или на 99% идентична последовательности с SEQ ID NO: 38, 40, 42, 44, 46, 48, 50, 52, 54, 56, 58, 60, 62, 64, 66, 68, 70, 72, 74, 76, 78, 80, 82, 84, 86, 88, 90, 92 или 94, или их соответствующим последовательностям РНК. В аспектах данного изобретения полинуклеотид, который образует комплекс со слитым белком, описанным в данном документе, включает в себя последовательность с SEQ ID NO: 38. В аспектах данного изобретения полинуклеотид, который образует комплекс со слитым белком, описанным в данном документе, включает в себя последовательность с SEQ ID NO: 40. В аспектах данного изобретения полинуклеотид, который образует комплекс со слитым белком, описанным в данном документе, включает в себя последовательность с SEQ ID NO: 42. В аспектах данного изобретения полинуклеотид, который образует комплекс со слитым белком, описанным в данном документе, включает в себя последовательность с SEQ ID NO: 44. В аспектах данного изобретения полинуклеотид, который образует комплекс со слитым белком, описанным в данном документе, включает в себя последовательность с SEQ ID NO: 46. В аспектах данного изобретения полинуклеотид, который образует комплекс со слитым белком, описанным в данном документе, включает в себя последовательность с SEQ ID NO: 48. В аспектах данного изобретения полинуклеотид, который образует комплекс со слитым белком, описанным в данном документе, включает в себя последовательность с SEQ ID NO: 50. В аспектах данного изобретения полинуклеотид, который образует комплекс со слитым белком, описанным в данном документе, включает в себя последовательность с SEQ ID NO: 52. В аспектах данного изобретения полинуклеотид, который образует комплекс со слитым белком, описанным в данном документе, включает в себя последовательность с SEQ ID NO: 54. В аспектах данного изобретения полинуклеотид, который образует комплекс со слитым белком, описанным в данном документе, включает в себя последовательность с SEQ ID NO: 56. В аспектах данного изобретения полинуклеотид, который образует комплекс со слитым белком, описанным в данном документе, включает в себя последовательность с SEQ ID NO: 58. В аспектах данного изобретения полинуклеотид, который образует комплекс со слитым белком, описанным в данном документе, включает в себя последовательность с SEQ ID NO: 60. В аспектах данного изобретения полинуклеотид, который образует комплекс со слитым белком, описанным в данном документе, включает в себя последовательность с SEQ ID NO: 62. В аспектах данного изобретения полинуклеотид, который образует комплекс со слитым белком, описанным в данном документе, включает в себя последовательность с SEQ ID NO: 64. В аспектах данного изобретения полинуклеотид, который образует комплекс со слитым белком, описанным в данном документе, включает в себя последовательность с SEQ ID NO: 66. В аспектах данного изобретения полинуклеотид, который образует комплекс со слитым белком, описанным в данном документе, включает в себя последовательность с SEQ ID NO: 68. В аспектах данного изобретения полинуклеотид, который образует комплекс со слитым белком, описанным в данном документе, включает в себя последовательность с SEQ ID NO: 70. В аспектах данного изобретения полинуклеотид, который образует комплекс со слитым белком, описанным в данном документе, включает в себя последовательность с SEQ ID NO: 72. В аспектах данного изобретения полинуклеотид, который образует комплекс со слитым белком, описанным в данном документе, включает в себя последовательность с SEQ ID NO: 74. В аспектах данного изобретения полинуклеотид, который образует комплекс со слитым белком, описанным в данном документе, включает в себя последовательность с SEQ ID NO: 76. В аспектах данного изобретения полинуклеотид, который образует комплекс со слитым белком, описанным в данном документе, включает в себя последовательность с SEQ ID NO: 78. В аспектах данного изобретения полинуклеотид, который образует комплекс со слитым белком, описанным в данном документе, включает в себя последовательность с SEQ ID NO: 80. В аспектах данного изобретения полинуклеотид, который образует комплекс со слитым белком, описанным в данном документе, включает в себя последовательность с SEQ ID NO: 82. В аспектах данного изобретения полинуклеотид, который образует комплекс со слитым белком, описанным в данном документе, включает в себя последовательность с SEQ ID NO: 84. В аспектах данного изобретения полинуклеотид, который образует комплекс со слитым белком, описанным в данном документе, включает в себя последовательность с SEQ ID NO: 86. В аспектах данного изобретения полинуклеотид, который образует комплекс со слитым белком, описанным в данном документе, включает в себя последовательность с SEQ ID NO: 88. В аспектах данного изобретения полинуклеотид, который образует комплекс со слитым белком, описанным в данном документе, включает в себя последовательность с SEQ ID NO: 90. В аспектах данного изобретения полинуклеотид, который образует комплекс со слитым белком, описанным в данном документе, включает в себя последовательность с SEQ ID NO: 92. В аспектах данного изобретения полинуклеотид, который образует комплекс со слитым белком, описанным в данном документе, включает в себя последовательность с SEQ ID NO: 94.
[0135] Нуклеиновые кислоты и векторы
[0136] Слитый белок, описанный в данном документе, включительно с его вариантами реализации и аспектами, может быть представлен в виде последовательности нуклеиновой кислоты, кодирующей слитый белок. Таким образом, в одном аспекте данного изобретения представлена последовательность нуклеиновой кислоты, кодирующая слитый белок, описанный в данном документе, включительно с его вариантами реализации и аспектами. В одном аспекте данного изобретения представлена последовательность нуклеиновой кислоты, кодирующая слитый белок, описанный в данном документе (включая с нацеливающую на ДНК последовательность), включительно с его вариантами реализации и аспектами. В аспектах данного изобретения последовательность нуклеиновой кислоты кодирует слитый белок, описанный в данном документе, включая слитые белки, имеющие аминокислотные последовательности с определенным % идентичности последовательностям, описанным в данном документе. В аспектах данного изобретения нуклеиновая кислота представляет собой РНК. В аспектах данного изобретения нуклеиновая кислота представляет собой информационную РНК. В аспектах данного изобретения информационная РНК представляет собой информационный РНП. В аспектах данного изобретения последовательность нуклеиновой кислоты кодирует слитые белки, описанные в данном документе, включительно с их вариантами реализации и аспектами. В аспектах данного изобретения последовательность нуклеиновой кислоты кодирует слитый белок с последовательностью SEQ ID NO: 1. В аспектах данного изобретения последовательность нуклеиновой кислоты кодирует слитый белок с последовательностью SEQ ID NO: 2. В аспектах данного изобретения последовательность нуклеиновой кислоты кодирует слитый белок с последовательностью SEQ ID NO: 3. В аспектах данного изобретения последовательность нуклеиновой кислоты кодирует слитый белок с последовательностью SEQ ID NO: 4. В аспектах данного изобретения последовательность нуклеиновой кислоты кодирует слитый белок с последовательностью SEQ ID NO: 5. В аспектах данного изобретения последовательность нуклеиновой кислоты кодирует слитый белок с последовательностью SEQ ID NO: 6. В аспектах данного изобретения последовательность нуклеиновой кислоты кодирует слитый белок с последовательностью SEQ ID NO: 7. В аспектах данного изобретения последовательность нуклеиновой кислоты кодирует слитый белок с последовательностью SEQ ID NO: 8. В аспектах данного изобретения последовательность нуклеиновой кислоты кодирует слитый белок с последовательностью SEQ ID NO: 9. В аспектах данного изобретения последовательность нуклеиновой кислоты кодирует слитый белок с последовательностью SEQ ID NO: 10. В аспектах данного изобретения последовательность нуклеиновой кислоты кодирует слитый белок с последовательностью SEQ ID NO: 11. В аспектах данного изобретения последовательность нуклеиновой кислоты кодирует слитый белок с последовательностью SEQ ID NO: 12. В аспектах данного изобретения последовательность нуклеиновой кислоты кодирует слитый белок с последовательностью SEQ ID NO: 13. В аспектах данного изобретения последовательность нуклеиновой кислоты кодирует слитый белок с последовательностью SEQ ID NO: 14. В аспектах данного изобретения последовательность нуклеиновой кислоты кодирует слитый белок с последовательностью SEQ ID NO: 15.
[0137] Кроме того, предполагается, что последовательность нуклеиновой кислоты, кодирующая слитый белок, как описано в данном документе, включительно с его вариантами реализации и аспектами, может быть включена в вектор. Таким образом, в одном аспекте данного изобретения представлен вектор, включающий в себя последовательность нуклеиновой кислоты, как описано в данном документе, включительно с его вариантами реализации и аспектами. В аспектах данного изобретения вектор содержит последовательность нуклеиновой кислоты, которая кодирует слитый белок, описанный в данном документе, включая слитые белки, имеющие аминокислотные последовательности с определенным % идентичности последовательностям, описанным в данном документе. В аспектах данного изобретения нуклеиновая кислота представляет собой информационную РНК. В аспектах данного изобретения информационная РНК представляет собой информационный РНП. В аспектах данного изобретения вектор содержит последовательность нуклеиновой кислоты, которая кодирует слитый белок с последовательностью SEQ ID NO: 1. В аспектах данного изобретения вектор содержит последовательность нуклеиновой кислоты, которая кодирует слитый белок с последовательностью SEQ ID NO: 2. В аспектах данного изобретения вектор содержит последовательность нуклеиновой кислоты, которая кодирует слитый белок с последовательностью SEQ ID NO: 3. В аспектах данного изобретения вектор содержит последовательность нуклеиновой кислоты, которая кодирует слитый белок с последовательностью SEQ ID NO: 4. В аспектах данного изобретения вектор содержит последовательность нуклеиновой кислоты, которая кодирует слитый белок с последовательностью SEQ ID NO: 5. В аспектах данного изобретения вектор содержит последовательность нуклеиновой кислоты, которая кодирует слитый белок с последовательностью SEQ ID NO: 6. В аспектах данного изобретения вектор содержит последовательность нуклеиновой кислоты, которая кодирует слитый белок с последовательностью SEQ ID NO: 7. В аспектах данного изобретения вектор содержит последовательность нуклеиновой кислоты, которая кодирует слитый белок с последовательностью SEQ ID NO: 8. В аспектах данного изобретения вектор содержит последовательность нуклеиновой кислоты, которая кодирует слитый белок с последовательностью SEQ ID NO: 9. В аспектах данного изобретения вектор содержит последовательность нуклеиновой кислоты, которая кодирует слитый белок с последовательностью SEQ ID NO: 10. В аспектах данного изобретения вектор содержит последовательность нуклеиновой кислоты, которая кодирует слитый белок с последовательностью SEQ ID NO: 11. В аспектах данного изобретения вектор содержит последовательность нуклеиновой кислоты, которая кодирует слитый белок с последовательностью SEQ ID NO: 12. В аспектах данного изобретения вектор содержит последовательность нуклеиновой кислоты, которая кодирует слитый белок с последовательностью SEQ ID NO: 13. В аспектах данного изобретения вектор содержит последовательность нуклеиновой кислоты, которая кодирует слитый белок с последовательностью SEQ ID NO: 14. В аспектах данного изобретения вектор содержит последовательность нуклеиновой кислоты, которая кодирует слитый белок с последовательностью SEQ ID NO: 15.
[0138] В вариантах реализации данного изобретения вектор дополнительно включает в себя полинуклеотид, при этом полинуклеотид включает в себя: (1) нацеливающую на ДНК последовательность, которая комплементарна целевой полинуклеотидной последовательности; и (2) связывающую последовательность для фермента нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК. Таким образом, один или несколько векторов могут включать в себя все необходимые компоненты для выполнения редактирования эпигенома.
[0139] Клетки
[0140] Композиции, описанные в данном документе, могут быть включены в клетку. Внутри клетки композиции, описанные в данном документе, включительно с их вариантами реализации и аспектами, могут выполнять редактирование эпигенома. Соответственно, в одном аспекте данного изобретения представлена клетка, которая включает в себя слитый белок, как описано в данном документе, включительно с его вариантами реализации и аспектами, нуклеиновую кислоту, как описано в данном документе, включительно с ее вариантами реализации и аспектами, комплекс, как описано в данном документе, включительно с его вариантами реализации и аспектами, или вектор, как описано в данном документе, включительно с его вариантами реализации и аспектами. В аспектах данного изобретения представлена клетка, которая включает в себя слитый белок, как описано в данном документе, включительно с его вариантами реализации и аспектами. В аспектах данного изобретения представлена клетка, которая включает в себя нуклеиновую кислоту, как описано в данном документе, включительно с ее вариантами реализации и аспектами. В аспектах данного изобретения представлена клетка, которая включает в себя комплекс, как описано в данном документе, включительно с его вариантами реализации и аспектами. В аспектах данного изобретения представлена клетка, которая включает в себя вектор, как описано в данном документе, включительно с его вариантами реализации и аспектами. В аспектах данного изобретения клетка представляет собой эукариотическую клетку. В аспектах данного изобретения клетка представляет собой клетку млекопитающего.
[0141] Способы
[0142] Предполагается, что композиции, описанные в данном документе, можно использовать для редактирования эпигенома, и более конкретно - для редактирования эпигенома, приводящего к репрессии или сайленсингу целевых последовательностей нуклеиновых кислот (например, генов). Без намерения быть связанными какой-либо теорией, сайленсинг может быть результатом метилирования и (или) введения репрессивных хроматиновых маркеров (например, моно-, ди- или триметилирование определенных гистонов (например, H3K9, H3K27), деацетилирование, ацетилирование, фосфорилирование, убиквитинирование) в хроматин, содержащий целевую последовательность нуклеиновой кислоты. Без намерения быть связанными какой-либо теорией, данный метод может быть использован для изменения эпигенетического состояния, например, путем закрытия хроматина посредством метилирования или введения репрессивных хроматиновых маркеров в хроматин, содержащий целевую последовательность нуклеиновой кислоты (например, ген). Без намерения быть связанными какой-либо теорией, предполагается, что слитый белок Dnmt3A-3L функционирует для добавления метильных меток в ДНК в сайтах CG, обнаруживаемых на островках CpG, а домен KRAB рекрутирует эпигенетические факторы, которые модифицируют гистоны путем введения репрессивных меток. Без намерения быть связанными какой-либо теорией, ДНК метилируется по нуклеотиду C в последовательностях CG, обнаруженных на островках CpG (т. е. добавляются метильные метки на нуклеотиде C в ДНК в сайтах CG, обнаруживаемых на островках CpG).
[0143] В одном аспекте данного изобретения представлен способ сайленсинга последовательности целевой нуклеиновой кислоты в клетке, включая доставку первого полинуклеотида, кодирующего слитый белок, как описано в данном документе, включительно с его вариантами реализации и аспектами, в клетку, содержащую целевую нуклеиновую кислоту; и доставку в клетку второго полинуклеотида, который включает в себя (i) нацеленную на ДНК последовательность, которая комплементарна последовательности целевой нуклеиновой кислоты, и (ii) связывающую последовательность для фермента нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК. Без намерения быть связанным какой-либо теорией, слитый белок вызывает сайленсинг последовательности целевой нуклеиновой кислоты в клетке путем метилирования хроматина, содержащего последовательность целевой нуклеиновой кислоты, и (или) путем введения репрессивных хроматиновых маркеров в хроматин, содержащий последовательность целевой нуклеиновой кислоты. Без намерения быть связанным какой-либо теорией, метилирование хроматина означает, что ДНК метилируется по нуклеотиду C в последовательностях CG, обнаруживаемых на островках CpG (т. е. добавляются метильные метки на нуклеотиде C в ДНК в сайтах CG, обнаруживаемых на островках CpG). В аспектах данного изобретения метилированной является последовательность, которая находится в пределах около 3000 пар оснований от последовательности целевой нуклеиновой кислоты. В аспектах данного изобретения метилированной является последовательность, которая находится в пределах около 3000, около 2900, около 2800, около 2700, около 2600, около 2500, около 2400, около 2300, около 2200, около 2100, около 2000, около 1900, около 1800, около 1700, около 1600, около 1500, около 1400, около 1300, около 1200, около 1100, около 1000, около 900, около 800, около 700, около 600, около 500, около 400, около 300, около 200 или около 100 пар оснований от последовательности целевой нуклеиновой кислоты.
[0144] Термин «репрессивные хроматиновые маркеры», употребляемый в контексте данного документа, относится к модификациям, внесенным в хроматин, которые приводят к сайленсингу (например, снижению или ингибированию транскрипции) последовательности целевой нуклеиновой кислоты (например, гена). Примеры репрессивных хроматиновых маркеров включают в себя, но не ограничиваются ими, моно-, ди- и (или) триметилирование, ацетилирование/ деацетилирование, фосфорилирование и убиквитинирование гистонов (например, H3K9, H3K27, H3K79, H2BK5).
[0145] В вариантах реализации данного изобретения сайленсинг относится к полному подавлению транскрипции. В аспектах данного изобретения сайленсинг относится к значительному снижению транскрипции по сравнению с контрольными уровнями транскрипции.
[0146] В вариантах реализации данного изобретения первый полинуклеотид содержится в первом векторе. В аспектах данного изобретения первый полинуклеотид содержится во втором векторе. В аспектах данного изобретения первый вектор и второй вектор одинаковы. В аспектах данного изобретения первый вектор отличается от второго вектора.
[0147] В вариантах реализации данного изобретения описанный в данном документе полинуклеотид доставляется в клетку любым способом, известным в данной области техники, например, путем трансфекции, электропорации или трансдукции.
[0148] Альтернативно, в одном аспекте данного изобретения представлен способ, приводящий к сайленсингу последовательности целевой нуклеиновой кислоты в клетке, включая доставку комплекса, как описано в данном документе, включительно с его вариантами реализации и аспектами, в клетку, которая содержит целевую нуклеиновую кислоту. Без намерения быть связанными какой-либо теорией, комплекс вызывает сайленсинг последовательности целевой нуклеиновой кислоты в клетке путем метилирования хроматина, содержащего последовательность целевой нуклеиновой кислоты, и (или) путем введения репрессивных хроматиновых маркеров в хроматин, содержащий последовательность целевой нуклеиновой кислоты.
[0149] В вариантах реализации данного изобретения клетка представляет собой клетку млекопитающего.
[0150] В вариантах реализации данного изобретения способ имеет специфичность, которая в 2 раза выше, чем специфичность к последовательности нецелевой нуклеиновой кислоты. В аспектах данного изобретения способ имеет специфичность, которая по меньшей мере в 2 раза (например, в 2, в 3, в 4, в 5, в 6, в 7, в 8, в 9, в 10, в 15, в 20, в 25 раз) выше, чем специфичность к последовательности нецелевой нуклеиновой кислоты. Методы определения специфичности хорошо известны в данной области техники и включают в себя, но не ограничиваются ими, секвенирование РНК, бисульфитное секвенирование, иммунопреципитацию хроматина, проточную цитометрию и количественную ПЦР. Таким образом, в аспектах данного изобретения специфичность определяется с помощью метода секвенирования РНК. В аспектах данного изобретения специфичность определяется с помощью метода бисульфитного секвенирования. В аспектах данного изобретения специфичность определяется с помощью метода иммунопреципитации хроматина. В аспектах данного изобретения специфичность определяется с помощью метода проточной цитометрии. В аспектах данного изобретения специфичность определяется с помощью метода количественной ПЦР.
[0151] В аспектах данного изобретения комплекс доставляется в клетку любыми способами, известными в данной области техники, например, посредством доставки рибонуклеопротеина (РНП).
[0152] Варианты реализации изобретения N1 - N41
[0153] Вариант реализации изобретения N1. Слитый белок, содержащий фермент нуклеазо-дефицитную РНК-направляемую эндонуклеазу ДНК, домен, связанный с боксом  , и домен метилтрансферазы ДНК.
, и домен метилтрансферазы ДНК.
[0154] Вариант реализации изобретения N2. Слитый белок по варианту реализации изобретения N1, отличающийся тем, что указанный фермент нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК представляет собой dCas9, ddCpf1, нуклеазо-дефицитный вариант Cas9 или нуклеазо-дефицитную эндонуклеазу CRISPR класса II.
[0155] Вариант реализации изобретения N3. Слитый белок по варианту реализации изобретения N1 или N2, отличающийся тем, что указанный домен метилтрансферазы ДНК представляет собой домен Dnmt3A-3L.
[0156] Вариант реализации изобретения N4. Слитый белок по варианту реализации изобретения N1, отличающийся тем, что указанный слитый белок включает в себя, в направлении от N-конца до С-конца, домен метилтрансферазы ДНК, фермент нуклеазо-дефицитную РНК-направляемую эндонуклеазу ДНК и домен, связанный с боксом  .
.
[0157] Вариант реализации изобретения N5. Слитый белок по варианту реализации изобретения N4, отличающийся тем, что указанный фермент нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК представляет собой dCas9, а указанный домен метилтрансферазы ДНК представляет собой домен Dnmt3A-3L.
[0158] Вариант реализации изобретения N6. Слитый белок по варианту реализации изобретения N5, отличающийся тем, что указанный фермент нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК представляет собой dCas9, а указанный домен метилтрансферазы ДНК представляет собой домен Dnmt3A-3L.
[0159] Вариант реализации изобретения N7. Слитый белок по варианту реализации изобретения N6, отличающийся тем, что указанный пептидный линкер представляет собой линкер XTEN.
[0160] Вариант реализации изобретения N8. Слитый белок по варианту реализации изобретения N1, отличающийся тем, что указанный слитый белок включает в себя, в направлении от N-конца до С-конца, домен, связанный с боксом  , фермент нуклеазо-дефицитную РНК-направляемую эндонуклеазу ДНК и домен метилтрансферазы ДНК.
, фермент нуклеазо-дефицитную РНК-направляемую эндонуклеазу ДНК и домен метилтрансферазы ДНК.
[0161] Вариант реализации изобретения N9. Слитый белок по варианту реализации изобретения N8, отличающийся тем, что указанный фермент нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК представляет собой dCas9, а указанный домен метилтрансферазы ДНК представляет собой домен Dnmt3A-3L.
[0162] Вариант реализации изобретения N10. Слитый белок по варианту реализации изобретения N9, отличающийся тем, что указанный dCas9 ковалентно связан с доменом Dnmt3A-3L через пептидный линкер, и при этом домен, связанный с боксом  , ковалентно связан с dCas9 через пептидный линкер.
, ковалентно связан с dCas9 через пептидный линкер.
[0163] Вариант реализации изобретения N11. Слитый белок по варианту реализации изобретения N10, отличающийся тем, что указанный пептидный линкер представляет собой линкер XTEN.
[0164] Вариант реализации изобретения N12. Слитый белок по любому из вариантов реализации изобретения с N1 по N3, отличающийся тем, что указанный фермент нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК ковалентно связан с доменом, связанным с боксом  , через пептидный линкер.
, через пептидный линкер.
[0165] Вариант реализации изобретения N13. Слитый белок по любому из вариантов реализации с N1 по N3, отличающийся тем, что указанный фермент нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК ковалентно связан с доменом метилтрансферазы ДНК через пептидный линкер.
[0166] Вариант реализации изобретения N14. Слитый белок по любому из вариантов реализации с N1 по N3, отличающийся тем, что указанный домен, связанный с боксом  , ковалентно связан с доменом метилтрансферазы ДНК через пептидный линкер.
, ковалентно связан с доменом метилтрансферазы ДНК через пептидный линкер.
[0167] Вариант реализации изобретения N15. Слитый белок по любому из вариантов реализации с N12 по N14, отличающийся тем, что указанный пептидный линкер представляет собой линкер XTEN.
[0168] Вариант реализации изобретения N16. Слитый белок по варианту реализации изобретения N15, отличающийся тем, что указанный линкер XTEN содержит от около 16 до около 80 аминокислотных остатков.
[0169] Вариант реализации изобретения N17. Слитый белок по любому из вариантов реализации с N1 по N16, дополнительно содержащий сигнальный пептид ядерной локализации.
[0170] Вариант реализации изобретения N18. Слитый белок по варианту реализации изобретения N1, отличающийся тем, что указанный слитый белок содержит аминокислотную последовательность с SEQ ID NO: 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14 или 15.
[0171] Вариант реализации изобретения N19. Последовательность нуклеиновой кислоты, кодирующая слитый белок по любому из вариантов реализации изобретения с N1 по N18.
[0172] Вариант реализации изобретения N20. Последовательность нуклеиновой кислоты по варианту реализации изобретения N19, отличающаяся тем, что указанная последовательность нуклеиновой кислоты представляет собой информационную РНК.
[0173] Вариант реализации изобретения N21. Комплекс, содержащий: (i) слитый белок по любому из вариантов реализации изобретения с N1 по N18; и (ii) полинуклеотид, содержащий: (а) нацеливающую на ДНК последовательность, комплементарную целевой полинуклеотидной последовательности; и (b) последовательность связывания для фермента нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК, при этом фермент нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК связан с полинуклеотидом через связывающую последовательность.
[0174] Вариант реализации изобретения N22. Комплекс по варианту реализации изобретения N21, отличающийся тем, что указанная целевая полинуклеотидная последовательность представляет собой часть гена.
[0175] Вариант реализации изобретения N23. Комплекс по варианту реализации изобретения N21, отличающийся тем, что указанная целевая полинуклеотидная последовательность представляет собой часть последовательности, регулирующей транскрипцию.
[0176] Вариант реализации изобретения N24. Комплекс по варианту реализации изобретения N21, отличающийся тем, что указанная целевая полинуклеотидная последовательность представляет собой часть промотора, энхансера или сайленсера.
[0177] Вариант реализации изобретения N25. Комплекс по варианту реализации изобретения N21, отличающийся тем, что указанная целевая полинуклеотидная последовательность находится в пределах около 3000 п.о. и фланкирует сайт начала транскрипции.
[0178] Вариант реализации изобретения N26. Вектор, содержащий последовательность нуклеиновой кислоты по варианту реализации изобретения N19 или N20.
[0179] Вариант реализации изобретения N27. Вектор по варианту реализации изобретения N26, дополнительно содержащий полинуклеотид, при этом полинуклеотид содержит: (а) нацеливающую на ДНК последовательность, которая комплементарна целевой полинуклеотидной последовательности; и (b) последовательность связывания для фермента нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК.
[0180] Вариант реализации изобретения N28. Клетка, содержащая слитый белок по любому из вариантов реализации изобретения с N1 по N18; нуклеиновую кислоту по варианту реализации изобретения N19 или N20; комплекс по любому из вариантов реализации изобретения с N21 по N25, или вектор по варианту реализации изобретения N26 или N27.
[0181] Вариант реализации изобретения N29. Клетка по варианту реализации изобретения N28, отличающаяся тем, что указанная клетка представляет собой эукариотическую клетку.
[0182] Вариант реализации изобретения N30. Клетка по варианту реализации изобретения N28, отличающаяся тем, что указанная клетка представляет собой клетку млекопитающего.
[0183] Вариант реализации изобретения N31. Способ сайленсинга последовательности целевой нуклеиновой кислоты в клетке, включающий: (i) доставку первого полинуклеотида, кодирующего слитый белок по любому из вариантов реализации изобретения с N1 по N18, в клетку, содержащую целевую нуклеиновую кислоту; и (ii) доставку в клетку второго полинуклеотида, который включает в себя (a) нацеленную на ДНК последовательность, которая комплементарна последовательности целевой нуклеиновой кислоты, и (b) связывающую последовательность для фермента нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК.
[0184] Вариант реализации изобретения N32. Способ по варианту реализации изобретения N31, отличающийся тем, что указанный слитый белок вызывает сайленсинг последовательности целевой нуклеиновой кислоты в клетке путем метилирования хроматина, содержащего последовательность целевой нуклеиновой кислоты, и (или) путем введения репрессивных хроматиновых маркеров в хроматин, содержащий последовательность целевой нуклеиновой кислоты.
[0185] Вариант реализации изобретения N33. Способ по варианту реализации изобретения N31 или N32, отличающийся тем, что указанный первый полинуклеотид содержится в первом векторе.
[0186] Вариант реализации изобретения N34. Способ по любому из вариантов реализации изобретения с N31 по N33, отличающийся тем, что указанный первый полинуклеотид содержится во втором векторе.
[0187] Вариант реализации изобретения N35. Способ по варианту реализации изобретения N34, отличающийся тем, что указанные первый вектор и второй вектор одинаковы.
[0188] Вариант реализации изобретения N36. Способ по варианту реализации изобретения N34, отличающийся тем, что указанный первый вектор отличается от второго вектора.
[0189] Вариант реализации изобретения N37. Способ по варианту реализации изобретения N31, отличающийся тем, что указанная клетка представляет собой клетку млекопитающего.
[0190] Вариант реализации изобретения N38. Способ по варианту реализации изобретения N31, отличающийся тем, что специфичность указанного способа в 2 раза выше, чем специфичность по отношению к последовательности нецелевой нуклеиновой кислоты.
[0191] Вариант реализации изобретения N39. Способ сайленсинга последовательности целевой нуклеиновой кислоты в клетке, включающий в себя доставку комплекса по любому из вариантов реализации изобретения с N21 по N25 в клетку, содержащую целевую нуклеиновую кислоту.
[0192] Вариант реализации изобретения N40. Способ по варианту реализации изобретения N39, отличающийся тем, что указанный комплекс вызывает сайленсинг последовательности целевой нуклеиновой кислоты в клетке путем метилирования хроматина, содержащего последовательность целевой нуклеиновой кислоты, и (или) путем введения репрессивных хроматиновых маркеров в хроматин, содержащий последовательность целевой нуклеиновой кислоты.
[0193] Вариант реализации изобретения N41. Способ по варианту реализации изобретения N39 или N40, отличающийся тем, что указанная клетка представляет собой клетку млекопитающего.
[0194] Вариант реализации изобретения N42. Способ по любому из вариантов реализации с N39 по N41, отличающийся тем, что специфичность указанного способа в 2 раза выше, чем специфичность по отношению к последовательности нецелевой нуклеиновой кислоты.
[0195] Варианты реализации изобретения с 1 по 36
[0196] Вариант реализации изобретения 1. Слитый белок, содержащий фермент нуклеазо-дефицитную РНК-направляемую эндонуклеазу ДНК, домен, связанный с боксом  (домен KRAB), и домен метилтрансферазы ДНК.
(домен KRAB), и домен метилтрансферазы ДНК.
[0197] Вариант реализации изобретения 2. Слитый белок по варианту реализации изобретения 1, отличающийся тем, что указанный фермент нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК представляет собой dCas9, ddCpf1, нуклеазо-дефицитный вариант Cas9 или нуклеазо-дефицитную эндонуклеазу CRISPR класса II.
[0198] Вариант реализации изобретения 3. Слитый белок по варианту реализации изобретения 1 или 2, отличающийся тем, что указанный домен метилтрансферазы ДНК представляет собой домен Dnmt3A-3L.
[0199] Вариант реализации изобретения 4. Слитый белок по любому из вариантов реализации изобретения с 1 по 3, отличающийся тем, что указанный фермент нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК ковалентно связан с доменом KRAB через пептидный линкер.
[0200] Вариант реализации изобретения 5. Слитый белок по любому из вариантов реализации изобретения с 1 по 4, отличающийся тем, что указанный фермент нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК ковалентно связан с доменом метилтрансферазы ДНК через пептидный линкер.
[0201] Вариант реализации изобретения 6. Слитый белок по любому из вариантов реализации изобретения с 1 по 5, отличающийся тем, что указанный домен KRAB ковалентно связан с доменом метилтрансферазы ДНК через пептидный линкер.
[0202] Вариант реализации изобретения 7. Слитый белок по любому из вариантов реализации изобретения с 4 по 6, отличающийся тем, что указанный пептидный линкер представляет собой линкер XTEN.
[0203] Вариант реализации изобретения 8. Слитый белок по варианту реализации изобретения 7, отличающийся тем, что указанный линкер XTEN содержит от около 16 до около 80 аминокислотных остатков.
[0204] Вариант реализации изобретения 9. Слитый белок по любому из вариантов реализации изобретения с 1 по 8, дополнительно содержащий сигнальный пептид ядерной локализации.
[0205] Вариант реализации изобретения 10. Слитый белок по любому из вариантов реализации изобретения с 1 по 9, отличающийся тем, что указанный слитый белок содержит, в направлении от N-конца до C-конца, домен KRAB, фермент нуклеазо-дефицитную РНК-направляемую эндонуклеазу ДНК, и домен метилтрансферазы ДНК.
[0206] Вариант реализации изобретения 11. Слитый белок по любому из вариантов реализации изобретения с 1 по 10, отличающийся тем, что указанный фермент нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК представляет собой dCas9, а указанный домен метилтрансферазы ДНК представляет собой домен Dnmt3A-3L.
[0207] Вариант реализации изобретения 12. Слитый белок по варианту реализации изобретения 11, отличающийся тем, что указанный dCas9 ковалентно связан с доменом KRAB через пептидный линкер, и при этом dCas9 ковалентно связан с доменом Dnmt3A-3L через пептидный линкер.
[0208] Вариант реализации изобретения 13. Слитый белок по варианту реализации изобретения 12, отличающийся тем, что указанный пептидный линкер представляет собой линкер XTEN.
[0209] Вариант реализации изобретения 14. Слитый белок по любому из вариантов реализации изобретения с 1 по 13, отличающийся тем, что указанный слитый белок содержит аминокислотную последовательность с SEQ ID NO: 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11 12, 13, 14 или 15.
[0210] Вариант реализации изобретения 15. Последовательность нуклеиновой кислоты, кодирующая слитый белок по любому из вариантов реализации изобретения с 1 по 14.
[0211] Вариант реализации изобретения 16. Комплекс, содержащий: (i) слитый белок по любому из вариантов реализации изобретения с 1по 18; и (ii) полинуклеотид, содержащий: (а) нацеливающую на ДНК последовательность, комплементарную целевой полинуклеотидной последовательности; и (b) последовательность связывания для фермента нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК, при этом фермент нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК связан с полинуклеотидом через связывающую последовательность.
[0212] Вариант реализации изобретения 17. Комплекс по варианту реализации изобретения 21, отличающийся тем, что указанная целевая полинуклеотидная последовательность представляет собой часть гена.
[0213] Вариант реализации изобретения 18. Комплекс по варианту реализации изобретения 21, отличающийся тем, что указанная целевая полинуклеотидная последовательность представляет собой часть последовательности, регулирующей транскрипцию.
[0214] Вариант реализации изобретения 19. Комплекс по варианту реализации изобретения 21, отличающийся тем, что указанная целевая полинуклеотидная последовательность представляет собой часть промотора, энхансера или сайленсера.
[0215] Вариант реализации изобретения 20. Комплекс по варианту реализации изобретения 21, отличающийся тем, что указанная целевая полинуклеотидная последовательность представляет собой гипометилированную последовательность нуклеиновой кислоты.
[0216] Вариант реализации изобретения 21. Комплекс по варианту реализации изобретения 21, отличающийся тем, что указанная целевая полинуклеотидная последовательность находится в пределах около 3000 п.о. и фланкирует сайт начала транскрипции.
[0217] Вариант реализации изобретения 22. Вектор, содержащий последовательность нуклеиновой кислоты по варианту реализации изобретения 19.
[0218] Вариант реализации изобретения 23. Вектор по варианту реализации изобретения 26, дополнительно содержащий полинуклеотид, при этом полинуклеотид содержит: (а) нацеливающую на ДНК последовательность, которая комплементарна целевой полинуклеотидной последовательности; и (b) последовательность связывания для фермента нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК.
[0219] Вариант реализации изобретения 24. Клетка, содержащая слитый белок по любому из вариантов реализации изобретения с 1 по 14; нуклеиновую кислоту по варианту реализации изобретения 15; комплекс по любому из вариантов реализации изобретения с 16 по 21; или вектор по варианту реализации изобретения 22 или 23.
[0220] Вариант реализации изобретения 25. Клетка по варианту реализации изобретения 28, отличающаяся тем, что указанная клетка представляет собой эукариотическую клетку.
[0221] Вариант реализации изобретения 26. Клетка по варианту реализации изобретения 28, отличающаяся тем, что указанная клетка представляет собой клетку млекопитающего.
[0222] Вариант реализации изобретения 27. Способ сайленсинга последовательности целевой нуклеиновой кислоты в клетке, включающий: (i) доставку первого полинуклеотида, кодирующего слитый белок по любому из вариантов реализации изобретения с 1 по 14, в клетку, содержащую целевую нуклеиновую кислоту; и (ii) доставку в клетку второго полинуклеотида, который включает в себя (a) нацеленную на ДНК последовательность, которая комплементарна последовательности целевой нуклеиновой кислоты, и (b) связывающую последовательность для фермента нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК, при этом указанный слитый белок вызывает сайленсинг последовательности целевой нуклеиновой кислоты в клетке путем метилирования хроматина, содержащего последовательность целевой нуклеиновой кислоты, и (или) путем введения репрессивных хроматиновых маркеров в хроматин, содержащий последовательность целевой нуклеиновой кислоты.
[0223] Вариант реализации изобретения 28. Способ по варианту реализации изобретения 27, отличающийся тем, что указанный первый полинуклеотид содержится в первом векторе.
[0224] Вариант реализации изобретения 29. Способ по варианту реализации изобретения 27, отличающийся тем, что указанный первый полинуклеотид содержится во втором векторе.
[0225] Вариант реализации изобретения 30. Способ по варианту реализации изобретения 28 или 29, отличающийся тем, что указанные первый вектор и второй вектор одинаковы.
[0226] Вариант реализации изобретения 31. Способ по варианту реализации изобретения 28 или 29, отличающийся тем, что указанный первый вектор отличается от второго вектора.
[0227] Вариант реализации изобретения 32. Способ по любому из вариантов реализации изобретения с 27 по 31, отличающийся тем, что клетка представляет собой клетку млекопитающего.
[0228] Вариант реализации изобретения 33. Способ по любому из вариантов реализации изобретения с 27 по 32, отличающийся тем, что специфичность указанного способа в 2 раза выше, чем специфичность по отношению к последовательности нецелевой нуклеиновой кислоты.
[0229] Вариант реализации изобретения 34. Способ сайленсинга последовательности целевой нуклеиновой кислоты в клетке, включающий в себя доставку комплекса по любому из вариантов реализации изобретения с 16 по 20 в клетку, содержащую целевую нуклеиновую кислоту, при этом комплекс вызывает сайленсинг последовательности целевой нуклеиновой кислоты в клетке посредством: (i) метилирования хроматина, содержащего последовательность целевой нуклеиновой кислоты, (ii) введения репрессивных хроматиновых маркеров в хроматин, содержащий последовательность целевой нуклеиновой кислоты, или (iii) метилирования хроматина, содержащего последовательность целевой нуклеиновой кислоты, и введения репрессивных хроматиновых маркеров в хроматин, содержащий последовательность целевой нуклеиновой кислоты.
[0230] Вариант реализации изобретения 35. Способ по варианту реализации изобретения 34, отличающийся тем, что указанная клетка представляет собой клетку млекопитающего.
[0231] Вариант реализации изобретения 36. Способ по варианту реализации изобретения 34 или 35, отличающийся тем, что специфичность указанного способа в 2 раза выше, чем специфичность по отношению к последовательности нецелевой нуклеиновой кислоты.
ПРИМЕРЫ
[0232] Варианты реализации изобретения и аспекты, указанные в данном документе, дополнительно проиллюстрированы следующими примерами. Примеры предназначены лишь для иллюстрации вариантов реализации изобретения и аспектов, и их не следует истолковывать как ограничение объема изобретения, указанного в данном документе.
[0233] Пример 1
[0234] Слитые с dCas9 эпигенетические модуляторы протестированы на предмет постоянного сайленсинга генов. Исходная версия (В1, p76 (SEQ ID NO: 1)) белка «все в одном» (Фиг. 1A) содержит домен KRAB, слитый с -N-концом dCas9 (SEQ ID NO: 23), отделенный линкером GGSGGGS (SEQ ID NO: 17), и Dnmt3A - Dnmt3L на С-конце dCas9 (отделенные линкером EASGSGRASPGIPGSTR (SEQ ID NO: 19)). Еще один белок «все в одном», который объединил домен KRAB (SEQ ID NO: 16), dCas9 (D10A, H208A), Dnmt3A - Dnmt3L (SEQ ID NO: 33; где SEQ ID NO: 26 представляет собой Dnmt3A, а SEQ ID NO: 28 представляет собой Dnmt3L) в один полипептид (Фиг. 1B). Со ссылкой на Фиг. 1B, белок dCas9 - KRAB был адаптирован из Gilbert et al., Cell 2013, для применения с технологией интерференции CRISPR (CRISPRi), а слитый белок dCas9 - Dnmt3A - Dnmt3L был адаптирован из Stepper et al., Nucleic Acids Research, 2016.
[0235] Активность эпигенетического редактора В1 была протестирована на клетках HEK293T с использованием репортера GFP, чувствительного к метилированию ДНК (адаптированного из Stelzer et al., Cell 2015), для оценки длительного сайленсинга, вызываемого белком «все в одном» (Фиг. 1C). Универсальный хроматинраскрывающий элемент (УХЭ, англ. «UCOE») добавляли перед островком GAPDH CpG (CGI) для того, чтобы предотвратить фоновый сайленсинг лентивирусного вектора в клетках млекопитающих. Ген gfp отключается при метилировании GAPDH CGI. A, B и C обозначают положения, которые мы кодировали единой гидовой РНК (егРНК) для нацеливания в промоторе. Эти целевые последовательности и соответствующие последовательности егРНК перечислены в Таблице 1 ниже. В клетки котрансфицировали две плазмиды, одна из которых кодировала мутантный белок «hit-and-run», а другая плазмида кодировала егРНК (Фиг. 1D). Через двое суток после трансфекции были отсортированы клетки, которые экспрессируют белок «hit-and-run» и вектор, экспрессирующий егРНК. Флуоресценцию GFP оценивали с течением времени с помощью метода проточной цитометрии. Популяцию клеток, подвергающихся длительному сайленсингу репортера GFP, наблюдали в случае, когда белок «все в одном» экспрессируется с егРНК (Фиг. 1E). Количество клеток, подвергающихся длительному сайленсингу, было выше, чем при воздействии dCas9 - Dnmt3A - Dnmt3L (без домена KRAB).
[0236] Таблица 1
(SEQ ID NO: 37)
(SEQ ID NO: 38)
(SEQ ID NO: 39)
(SEQ ID NO: 40)
(SEQ ID NO: 41)
(SEQ ID NO: 42)
[0237] Сайленсинг репортера GFP зависит от последовательности егРНК, при этом гидовая последовательность C вызывает самый высокий уровень сайленсинга среди трех протестированных последовательностей егРНК. Объединение егРНК, кодирующих различные последовательности, в единый пул не привело к значительному изменению в сайленсинге генов.
[0238] Пример 2
[0239] Три гена (CD29, CD81, CD151) были мишенями для долгосрочного сайленсинга с использованием слитого белка «hit-and-run». Все три белка локализованы на поверхности клетки, и нокдаун оценивали путем окрашивания клеток антителами на поверхности клетки с последующей проточной цитометрией. Репрезентативные данные проточной цитометрии, полученные через 22 суток после трансфекции, показаны на Фиг. 2A - 2C. Квадрант IV представляет клетки, которые отключили ген, на что указывает процент клеток с отключенным геном. Отсутствие клеток в квадрантах I и II означает, что белок «hit-and-run» (содержащий метку СФБ) больше не присутствует в клетках. На Фиг. 2D представлена количественная оценка сайленсинга генов CD29, CD81 и CD151 с помощью трех различных егРНК, вызванного с помощью трех различных последовательностей егРНК или пула всех трех егРНК. Целевые последовательности ДНК и их егРНК, используемые в этом эксперименте, обобщены в Таблице 2.
[0240] Таблица 2
(SEQ ID NO: 43)
(SEQ ID NO: 44)
(SEQ ID NO: 46)
(SEQ ID NO: 48)
(SEQ ID NO: 49)
(SEQ ID NO: 50)
(SEQ ID NO: 52)
(SEQ ID NO: 54)
(SEQ ID NO: 56)
(SEQ ID NO: 57)
(SEQ ID NO: 58)
(SEQ ID NO: 59)
(SEQ ID NO: 60)
[0241] Одновременно мишенями были два или три гена для того, чтобы показать, что белок «все в одном» может быть мультиплексирован путем совместной доставки егРНК, нацеленных на разные гены. НК егРНК относится к ненацеленному контролю егРНК. Результаты показаны на Фиг. 2E.
[0242] Наблюдался генный сайленсинг в клетках, которые берут свое начало как единичный клон, и в большинстве клеток целевой ген CLTA оставался выключенным (37 из 39 клонов). График на Фиг. 2F представляет собой момент времени, взятый через 9 месяцев после трансфекции белка «все в одном» и егРНК, нацеленной на ген CLTA.
[0243] Описанная в данном документе система может нацеливаться на любые гены в геномах млекопитающих, особенно на те, которые содержат островки CpG на промоторе гена. Dnmt3A - Dnmt3L канонически нацелен на динуклеотиды CpG. Примеры генов, на которые может быть нацелена указанная система, включают в себя, но не ограничиваются ими, CXCR4, CD4, CD8, CD45, PD-1, CLTA-4, TGFBR, TCRa, TCRb, B2M.
[0244] Пример 3
[0245] Собирали клетки, которые утратили экспрессию ITGB1 (CD29), CD81 и CD151 через тридцать шесть суток после трансфекции, и анализировали их профили экспрессии РНК. Как показано на Фиг. 3A - 3C, был обнаружен успешный нокдаун целевых генов по сравнению с ненацеленным контролем егРНК. Фиг. 3D - 3F представляют собой вулканные диаграммы, показывающие, что нокдаун целевого гена является единственным значимым генным нокдауном для каждого эксперимента, что свидетельствует о высокой специфичности генного сайленсинга. Фиг. 3G - 3I представляют собой количественную оценку уровней транскриптов, показывающая более чем 96%-й нокдаун целевого гена.
[0246] Пример 4
[0247] Белок «все в одном» можно трансфицировать и экспрессировать в линии HeLa (клетки шейки матки), U2OS (клетки кости) и индуцированных плюрипотентных стволовых клетках человека (ИПСК). Графики проточной цитометрии на Фиг. 4A - 4F показывают экспрессию СФБ, который слит с белком. Мишенями были три эндогенных гена в клетках HeLa и U2OS (т. е. CD29, CD81 и CD151). Как показано на Фиг. 4G, был обнаружен стабильный сайленсинг, измеренный через 18 суток после трансфекции. При нацеливании на Pcsk9, Npc1, Spcs1 и Cd81 был обнаружен сайленсинг генов в клеточных линиях гепатоцитов мышей AML12. Сайленсинг был обнаружен с помощью кПЦР через 14 суток после трансфекции, как показано на Фиг. 4H. Последовательности егРНК, использованные в данном эксперименте, приведены в Таблице 3.
[0248] Таблица 3
(от 5' к 3')
(SEQ ID NO: 43)
(SEQ ID NO: 44)
(SEQ ID NO: 61)
(SEQ ID NO: 62)
(SEQ ID NO: 63)
(SEQ ID NO: 64)
(SEQ ID NO: 65)
(SEQ ID NO: 66)
(SEQ ID NO: 67)
(SEQ ID NO: 68)
(SEQ ID NO: 69)
(SEQ ID NO: 70)
(SEQ ID NO: 71)
(SEQ ID NO: 72)
(SEQ ID NO: 73)
(SEQ ID NO: 74)
(SEQ ID NO: 75)
(SEQ ID NO: 76)
(SEQ ID NO: 77)
(SEQ ID NO: 78)
(SEQ ID NO: 79)
(SEQ ID NO: 80)
(SEQ ID NO: 81)
(SEQ ID NO: 82)
(SEQ ID NO: 60)
(SEQ ID NO: 60)
(SEQ ID NO: 83)
(SEQ ID NO: 84)
(SEQ ID NO: 85)
(SEQ ID NO: 86)
(SEQ ID NO: 87)
(SEQ ID NO: 88)
(SEQ ID NO: 89)
(SEQ ID NO: 90)
(SEQ ID NO: 91)
(SEQ ID NO: 92)
(SEQ ID NO: 93)
(SEQ ID NO: 94)
(SEQ ID NO: 95)
(SEQ ID NO: 96)
[0249] Пример 5
[0250] На Фиг. 5 представлена схема белковых конструкций «все в одном», которые были сконструированы и протестированы на предмет сайленсинга генов. Первоначальный формат (p76, В1) SEQ ID NO: 1 был модифицирован для кодирования линкеров XTEN (например, 16 аминокислот (SEQ ID NO: 31) или 80 аминокислот (SEQ ID NO: 32)) либо на N-конце, либо на С-конце dCas9 (SEQ ID NO: 29). Все векторы содержат метки HA (SEQ ID NO: 24) на С-конце dCas9. В аспектах данного изобретения используется промотор CAG, поскольку он обеспечивает хорошую экспрессию, например, в конструкциях p76 и p90-102, p112 (В2). Со ссылкой на Фиг. 5, белковые конструкции с p90 по p102 соответствуют последовательностям с SEQ ID NO: 2-14, соответственно, а белковые конструкции p112 соответствуют последовательности с SEQ ID NO: 15.
[0251] Пример 6
[0252] Белковые конструкции, показанные на Фиг. 5, тестировали на предмет сайленсинга гена CLTA в клетках HEK293T в течение 18 суток после трансфекции (Фиг. 6A - 6B). Были обнаружены переменные уровни активности в сайленсинге генов, включая ряд вариантов с более устойчивым сайленсингом генов по сравнению с конструкцией p76 (В1), таких как p99 (SEQ ID NO: 11), p100 (SEQ ID NO: 12) и p112 (SEQ ID NO: 15). На Фиг. 6A и 6B показано, что конструкции dCas9 - KRAB и dCas9 - Dnmt3A - Dnmt3L продемонстрировали кратковременную и более низкую эффективность длительного сайленсинга.
[0253] p76 (SEQ ID NO: 1), p112 (SEQ ID NO: 15) тестировали на предмет сайленсинга эндогенного гена HIST2H2BE (H2B) и синтетического репортерного гена Snrpn-GFP, стабильно экспрессируемого в клетках HEK293T (Фиг. 6C - 6D). За клетками наблюдали в течение 50 суток после трансфекции. Вариант p112 поддерживал сайленсинг гена с более высокой эффективностью, чем конструкция p76 (В1). Слитые белки dCas9 - Dnmt3A - Dnmt3L и dCas9 - KRAB продемонстрировали кратковременную и более низкую эффективность длительного сайленсинга. На Фиг. 6E представлен график экспрессии белков p76 и p112 в течение 50 суток для выключения гена HIST2H2BE (H2B). Уровни белка измеряли с помощью метода проточной цитометрии для детектирования СФБ, который экспрессируется совместно с белком «все в одном».
[0254] Пример 7
[0255] Анализ по методу вестерн-блоттинга проводили с вариантами белков «все в одном» p76, p90 - p102 с использованием антитела против Steptococcus pyogenes Cas9. Со ссылкой на Фиг. 7А, верхняя полоса представляет собой полноразмерный белок, а полосы меньшего размера представляют собой продукты протеолиза белка «все в одном». Варианты, для которых наблюдается слабый протеолиз, такие как p99 (SEQ ID NO: 11), p100 (SEQ ID NO: 12) и p102 (SEQ ID NO: 14), демонстрируют более высокую эффективность генного сайленсинга. Варианты с высокими уровнями протеолиза, такие как p96 (SEQ ID NO: 8) и p97 (SEQ ID NO: 9), приводили к более низкой эффективности устойчивого генного сайленсинга.
[0256] Анализ по методу вестерн-блоттинга проводили с вариантами белков «все в одном» для обнаружения свободного Dnmt3A, который отщепляется от слитого белка. Как показано на Фиг. 7B, варианты, для которых наблюдался слабо детектируемый свободный Dnmt3 или он вообще отсутствовал, такие как p92 (SEQ ID NO: 4), p100 (SEQ ID NO: 12), p101 (SEQ ID NO: 13) и p102 (SEQ ID NO: 14), проявляли более высокую эффективность устойчивого генного сайленсинга по сравнению с вариантами с детектируемым отщепленным Dnmt3A, то есть p76 (SEQ ID NO: 1), p91 (SEQ ID NO: 3), p96 (SEQ ID NO: 8), p98 (SEQ ID NO: 10).
[0257] Пример 8
[0258] Был проведен объединенный скрининг, как показано на Фиг. 8A, с целью определения оптимальных егРНК, которые приводят к долгосрочному сайленсингу генов. Использовали четыре клеточные линии HEK293T, каждая с различным геном с меткой GFP (CLTA, VIM, HIST2H2BE (H2B) и RAB11A). Тайлинг-библиотеки, состоящие из егРНК, охватывающих +/- 2,5 т.п.о. от сайта начала транскрипции (СНТ) каждого гена, стабильно экспрессировались в клетках путем доставки лентивирусов с последующей кратковременной экспрессией плазмидной ДНК, экспрессирующей белок «все в одном». Через четыре недели после трансфекции клетки, которые поддерживали сайленсинг генов, сортировали для идентификации конкретных егРНК. Фиг. 8B - 8E представляют собой гистограммы проточной цитометрии, показывающие процент клеток, подвергающихся генному сайленсингу через четыре недели после трансфекции.
[0259] Фиг. 9A - 9D представляют собой карты функциональности егРНК в сайте начала транскрипции целевого гена (CLTA, H2B, RAB11, VIM). Сайт начала транскрипции (СНТ, англ. «TSS») и островок CpG аннотированы над каждым графиком. Каждая точка отображает собой одну егРНК, а ее эффективность в долгосрочном генном сайленсинге отображено как изменение в log2-кратном содержании егРНК. Степень заполнения нуклеосом (нижний график) нанесена на графики по сигналу от МНазы (микрококковая нуклеаза, англ. «MNase»).
[0260] Пример 9
[0261] На Фиг. 10А представлен рабочий процесс объединенного скрининга в клетках HEK293T, проводимого с целью определения оптимальных положений нацеливания егРНК для белка «все в одном», адаптированный из предыдущего рицинового скрининга на биочипах высокой плотности в клетках K562 для определения оптимальных егРНК для dCas9 - KRAB (Gilbert, Horlbeck et al., Cell 2014). Сначала егРНК стабильно экспрессировали в клетках HEK293T путем доставки лентивирусов с последующей кратковременной трансфекцией плазмиды, кодирующей белок «все в одном» (День 0). Клетки, экспрессирующие белок «все в одном», сортировали (день 2) и позволяли им расти далее в течение трех суток. На 5-е сутки клетки рассаживали, и половину из них отбирали как образцы начального момента времени, а другую половину культивировали в течение еще десяти суток (День 15) как образцы конечного момента времени. Ростовой фенотип (γ) рассчитывается как log2 обогащения егРНК, деленный на количество удвоений клеток между T (начальное) и T (конечное).
[0262] На Фиг. 10B - 10E представлены репрезентативные графики, демонстрирующие ростовые фенотипы для четырех генов (ARL1, EIF6, SMC3, HEATR1) из существующих наборов данных dCas9 - KRAB/ CRISPRi в клетках K562 (Gilbert, Horlbeck et al., 2014) и с белком «все в одном» (нижний график). Каждая точка отображает собой егРНК. СНТ и аннотированный островок CpG показаны для каждого гена. Функциональные егРНК, использующие белок «все в одном», охватывают более широкий диапазон, чем просто функциональные егРНК, что означает более широкий диапазон эффективного нацеливания.
[0263] Пример 10
[0264] На Фиг. 11A - 11B представлено сравнение ростовых фенотипов и позиционирования нуклеосом (по сигналу от МНазы) для VPS53 и VPS54, и показано расположение функциональных егРНК в областях, лишенных нуклеосом. Более того, диапазон функциональных егРНК является более широким при использовании белка «все в одном» по сравнению с dCas9 - KRAB/ CRISPRi.
[0265] Пример 11
[0266] Транскрипция in vitro двух вариантов «все в одном» (p102 (SEQ ID NO: 14) и p112 (SEQ ID NO: 15)) демонстрирует полноразмерный синтез каждой конструкции (Фиг. 12A). На Фиг. 12B представлен график проточной цитометрии, показывающий экспрессию p102 и p112 через одни сутки после трансфекции мРНК в клетки HEK293T. На Фиг. 12C показана сайленсинг эндогенного гена CLTA с течением времени в клетках HEK293T после трансфекции мРНК, экспрессирующей варианты p102 и p112 белка «все в одном».
[0267] Пример 12
[0268] На Фиг. 13A представлены графики проточной цитометрии, показывающие индуцированную экспрессию белка «все в одном», полученную путем добавления доксициклина в клетки K562, которые стабильно кодируют белок «все в одном» под действием промотора, индуцируемого доксициклином. Экспрессию белка наблюдали в течение четырех суток после индукции доксициклином. Пунктирные линии на панелях на Фиг. 13А представляют собой базовую медианную флуоресценцию СФБ без введения доксициклина. Проводили вестерн-блоттинг клеток с целью выявления экспрессии белка «все в одном» до и после обработки доксициклином (Фиг. 13B). Присутствие белка «все в одном» не детектируется через 96 часов после индукции. Графики проточной цитометрии нокдауна CD81 и CD151 через 14 суток после обработки клеток K562 доксициклином представлены на Фиг. 13C - 13F. Показан процент клеток с отключенным целевым геном. Детектируемая экспрессия белка «все в одном» отсутствует, поскольку в квадрантах BFP+ нет клеток. Количественная оценка нокдауна CD81 и CD151 через 14 суток после обработки доксициклином или без обработки доксициклином представлена на Фиг. 13G.
[0269] Список литературы
[0270] Ecco et al, Development 144, 2017. Lambert et al., Cell 172, 2018. Siddique et al., J. Mol. Biol., 425, 2013. Stepper et al., Nucleic Acids Res., 45, 2017. Shmakov et al., Nat. Rev. Microbiol. 15, 2017. CCebrian-Serrano et al., Mamm. Genome 7-8, 2017. Pulecio et al., Cell Stem Cell 21, 2017.
[0271] Неофициальный список последовательностей
[0272] SEQ ID NO: 1 (p76 (последовательность белка «все в одном», версия 1): KRAB (жирный шрифт; из Gilbert et al., Cell, 2013, 2014), линкеры (подчеркивание), dCas9 (курсив), метка HA (строчные буквы), SV40 СЯЛ (строчные буквы, курсив), Dnmt3A (жирный шрифт, курсив; остатки 612-912; из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), линкер из 27 аминокислот (подчеркивание, курсив; из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), Dnmt3L (подчеркивание, жирный шрифт; из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), последовательность расщепления пептидом P2A (строчные буквы, жирный шрифт), СФБ (строчные буквы, подчеркивание))
DAKSLTAWSRTLVTFKDVFVDFTREEWKLLDTAQQIVYRNVMLENYKNLVSLGYQLTKPDVILRLEKGEEP GGSGGGS MDKKYSIGLAIGTNSVGWAVITDEYKVPSKKFKVLGNTDRHSIKKNLIGALLFDSGETAEATRLKRTARRRYTRRKNRICYLQEIFSNEMAKVDDSFFHRLEESFLVEEDKKHERHPIFGNIVDEVAYHEKYPTIYHLRKKLVDSTDKADLRLIYLALAHMIKFRGHFLIEGDLNPDNSDVDKLFIQLVQTYNQLFEENPINASGVDAKAILSARLSKSRRLENLIAQLPGEKKNGLFGNLIALSLGLTPNFKSNFDLAEDAKLQLSKDTYDDDLDNLLAQIGDQYADLFLAAKNLSDAILLSDILRVNTEITKAPLSASMIKRYDEHHQDLTLLKALVRQQLPEKYKEIFFDQSKNGYAGYIDGGASQEEFYKFIKPILEKMDGTEELLVKLNREDLLRKQRTFDNGSIPHQIHLGELHAILRRQEDFYPFLKDNREKIEKILTFRIPYYVGPLARGNSRFAWMTRKSEETITPWNFEEVVDKGASAQSFIERMTNFDKNLPNEKVLPKHSLLYEYFTVYNELTKVKYVTEGMRKPAFLSGEQKKAIVDLLFKTNRKVTVKQLKEDYFKKIECFDSVEISGVEDRFNASLGTYHDLLKIIKDKDFLDNEENEDILEDIVLTLTLFEDREMIEERLKTYAHLFDDKVMKQLKRRRYTGWGRLSRKLINGIRDKQSGKTILDFLKSDGFANRNFMQLIHDDSLTFKEDIQKAQVSGQGDSLHEHIANLAGSPAIKKGILQTVKVVDELVKVMGRHKPENIVIEMARENQTTQKGQKNSRERMKRIEEGIKELGSQILKEHPVENTQLQNEKLYLYYLQNGRDMYVDQELDINRLSDYDVDAIVPQSFLKDDSIDNKVLTRSDKNRGKSDNVPSEEVVKKMKNYWRQLLNAKLITQRKFDNLTKAERGGLSELDKAGFIKRQLVETRQITKHVAQILDSRMNTKYDENDKLIREVKVITLKSKLVSDFRKDFQFYKVREINNYHHAHDAYLNAVVGTALIKKYPKLESEFVYGDYKVYDVRKMIAKSEQEIGKATAKYFFYSNIMNFFKTEITLANGEIRKRPLIETNGETGEIVWDKGRDFATVRKVLSMPQVNIVKKTEVQTGGFSKESILPKRNSDKLIARKKDWDPKKYGGFDSPTVAYSVLVVAKVEKGKSKKLKSVKELLGITIMERSSFEKNPIDFLEAKGYKEVKKDLIIKLPKYSLFELENGRKRMLASAGELQKGNELALPSKYVNFLYLASHYEKLKGSPEDNEQKQLFVEQHKHYLDEIIEQISEFSKRVILADANLDKVLSAYNKHRDKPIREQAENIIHLFTLTNLGAPAAFKYFDTTIDRKRYTSTKEVLDATLIHQSITGLYETRIDLSQLGGD SRADypydvpdyaSGSpkkkrkvEASGSGRASPGIPGSTRNHDQEFDPPKVYPPVPAEKRKPIRVLSLFDGIATGLLVLKDLGIQVDRYIASEVCEDSITVGMVRHQGKIMYVGDVRSVTQKHIQEWGPFDLVIGGSPCNDLSIVNPARKGLYEGTGRLFFEFYRLLHDARPKEGDDRPFFWLFENVVAMGVSDKRDISRFLESNPVMIDAKEVSAAHRARYFWGNLPGMNRPLASTVNDKLELQECLEHGRIAKFSKVRTITTRSNSIKQGKDQHFPVFMNEKEDILWCTEMERVFGFPVHYTDVSNMSRLARQRLLGRSWSVPVIRHLFAPLKEYFACVSSGNSNANSRGPSFSSGLVPLSLRGSHMGPMEIYKTVSAWKRQPVRVLSLFRNIDKVLKSLGFLESGSGSGGGTLKYVEDVTNVVRRDVEKWGPFDLVYGSTQPLGSSCDRCPGWYMFQFHRILQYALPRQESQRPFFWIFMDNLLLTEDDQETTTRFLQTEAVTLQDVRGRDYQNAMRVWSNIPGLKSKHAPLTPKEEEYLQAQVRSRSKLDAPKVDLLVKNCLLPLREYFKYFSQNSLPLSRADpkkkrkvGSGatnfsllkqagdveenpgpselikenmhmklymegtvdnhhfkctsegegkpyegtqtmrikvveggplpfafdilatsflygsktfinhtqgipdffkqsfpegftwervttyedggvltatqdtslqdgcliynvkirgvnftsngpvmqkktlgweaftetlypadgglegrndmalklvggshlianikttyrskkpaknlkmpgvyyvdyrlerikeannetyveqhevavarycdlpsklghkln*
[0273] SEQ ID NO: 2 (p90 (KRAB - dCas9 - XTEN16 - Dnmt3A - Dnmt3L - P2A - BFP): KRAB (жирный шрифт, из Gilbert et al., Cell, 2013, 2014); линкеры (подчеркивание), dCas9 (курсив); метка HA (строчные буквы), SV40 СЯЛ (строчные буквы, курсив), XTEN16 (заглавные буквы, последовательность из 16 аминокислот), Dnmt3A (жирный шрифт, курсив; из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), линкер из 27 аминокислот (подчеркивание, курсив, из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), Dnmt3L (подчеркивание, жирный шрифт, из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), последовательность расщепления пептидом P2A (строчные буквы, жирный шрифт), BFP (строчные буквы, подчеркивание))
DAKSLTAWSRTLVTFKDVFVDFTREEWKLLDTAQQIVYRNVMLENYKNLVSLGYQLTKPDVILRLEKGEEP GGSGGGS MDKKYSIGLAIGTNSVGWAVITDEYKVPSKKFKVLGNTDRHSIKKNLIGALLFDSGETAEATRLKRTARRRYTRRKNRICYLQEIFSNEMAKVDDSFFHRLEESFLVEEDKKHERHPIFGNIVDEVAYHEKYPTIYHLRKKLVDSTDKADLRLIYLALAHMIKFRGHFLIEGDLNPDNSDVDKLFIQLVQTYNQLFEENPINASGVDAKAILSARLSKSRRLENLIAQLPGEKKNGLFGNLIALSLGLTPNFKSNFDLAEDAKLQLSKDTYDDDLDNLLAQIGDQYADLFLAAKNLSDAILLSDILRVNTEITKAPLSASMIKRYDEHHQDLTLLKALVRQQLPEKYKEIFFDQSKNGYAGYIDGGASQEEFYKFIKPILEKMDGTEELLVKLNREDLLRKQRTFDNGSIPHQIHLGELHAILRRQEDFYPFLKDNREKIEKILTFRIPYYVGPLARGNSRFAWMTRKSEETITPWNFEEVVDKGASAQSFIERMTNFDKNLPNEKVLPKHSLLYEYFTVYNELTKVKYVTEGMRKPAFLSGEQKKAIVDLLFKTNRKVTVKQLKEDYFKKIECFDSVEISGVEDRFNASLGTYHDLLKIIKDKDFLDNEENEDILEDIVLTLTLFEDREMIEERLKTYAHLFDDKVMKQLKRRRYTGWGRLSRKLINGIRDKQSGKTILDFLKSDGFANRNFMQLIHDDSLTFKEDIQKAQVSGQGDSLHEHIANLAGSPAIKKGILQTVKVVDELVKVMGRHKPENIVIEMARENQTTQKGQKNSRERMKRIEEGIKELGSQILKEHPVENTQLQNEKLYLYYLQNGRDMYVDQELDINRLSDYDVDAIVPQSFLKDDSIDNKVLTRSDKNRGKSDNVPSEEVVKKMKNYWRQLLNAKLITQRKFDNLTKAERGGLSELDKAGFIKRQLVETRQITKHVAQILDSRMNTKYDENDKLIREVKVITLKSKLVSDFRKDFQFYKVREINNYHHAHDAYLNAVVGTALIKKYPKLESEFVYGDYKVYDVRKMIAKSEQEIGKATAKYFFYSNIMNFFKTEITLANGEIRKRPLIETNGETGEIVWDKGRDFATVRKVLSMPQVNIVKKTEVQTGGFSKESILPKRNSDKLIARKKDWDPKKYGGFDSPTVAYSVLVVAKVEKGKSKKLKSVKELLGITIMERSSFEKNPIDFLEAKGYKEVKKDLIIKLPKYSLFELENGRKRMLASAGELQKGNELALPSKYVNFLYLASHYEKLKGSPEDNEQKQLFVEQHKHYLDEIIEQISEFSKRVILADANLDKVLSAYNKHRDKPIREQAENIIHLFTLTNLGAPAAFKYFDTTIDRKRYTSTKEVLDATLIHQSITGLYETRIDLSQLGGD SRADypydvpdyaSGSpkkkrkvSPGSGSETPGTSESATPESNHDQEFDPPKVYPPVPAEKRKPIRVLSLFDGIATGLLVLKDLGIQVDRYIASEVCEDSITVGMVRHQGKIMYVGDVRSVTQKHIQEWGPFDLVIGGSPCNDLSIVNPARKGLYEGTGRLFFEFYRLLHDARPKEGDDRPFFWLFENVVAMGVSDKRDISRFLESNPVMIDAKEVSAAHRARYFWGNLPGMNRPLASTVNDKLELQECLEHGRIAKFSKVRTITTRSNSIKQGKDQHFPVFMNEKEDILWCTEMERVFGFPVHYTDVSNMSRLARQRLLGRSWSVPVIRHLFAPLKEYFACVSSGNSNANSRGPSFSSGLVPLSLRGSHMGPMEIYKTVSAWKRQPVRVLSLFRNIDKVLKSLGFLESGSGSGGGTLKYVEDVTNVVRRDVEKWGPFDLVYGSTQPLGSSCDRCPGWYMFQFHRILQYALPRQESQRPFFWIFMDNLLLTEDDQETTTRFLQTEAVTLQDVRGRDYQNAMRVWSNIPGLKSKHAPLTPKEEEYLQAQVRSRSKLDAPKVDLLVKNCLLPLREYFKYFSQNSLPLSRADpkkkrkvGSGatnfsllkqagdveenpgpselikenmhmklymegtvdnhhfkctsegegkpyegtqtmrikvveggplpfafdilatsflygsktfinhtqgipdffkqsfpegftwervttyedggvltatqdtslqdgcliynvkirgvnftsngpvmqkktlgweaftetlypadgglegrndmalklvggshlianikttyrskkpaknlkmpgvyyvdyrlerikeannetyveqhevavarycdlpsklghkln *
[0274] SEQ ID NO: 3 (p91 (KRAB - dCas9 - Dnmt3A - Dnmt3L - P2A - P2A - BFP): KRAB (жирный шрифт, из Gilbert et al., Cell, 2013, 2014), линкеры (подчеркивание), dCas9 (курсив), метка HA (строчные буквы), SV40 СЯЛ (строчные буквы, курсив), Dnmt3A (жирный шрифт, курсив, из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), линкер из 27 аминокислот (подчеркивание, курсив, из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), Dnmt3L (подчеркивание, жирный шрифт, из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), последовательность расщепления пептидом P2A (строчные буквы, жирный шрифт), BFP (строчные буквы, подчеркивание))
DAKSLTAWSRTLVTFKDVFVDFTREEWKLLDTAQQIVYRNVMLENYKNLVSLGYQLTKPDVILRLEKGEEP GGSGGGS MDKKYSIGLAIGTNSVGWAVITDEYKVPSKKFKVLGNTDRHSIKKNLIGALLFDSGETAEATRLKRTARRRYTRRKNRICYLQEIFSNEMAKVDDSFFHRLEESFLVEEDKKHERHPIFGNIVDEVAYHEKYPTIYHLRKKLVDSTDKADLRLIYLALAHMIKFRGHFLIEGDLNPDNSDVDKLFIQLVQTYNQLFEENPINASGVDAKAILSARLSKSRRLENLIAQLPGEKKNGLFGNLIALSLGLTPNFKSNFDLAEDAKLQLSKDTYDDDLDNLLAQIGDQYADLFLAAKNLSDAILLSDILRVNTEITKAPLSASMIKRYDEHHQDLTLLKALVRQQLPEKYKEIFFDQSKNGYAGYIDGGASQEEFYKFIKPILEKMDGTEELLVKLNREDLLRKQRTFDNGSIPHQIHLGELHAILRRQEDFYPFLKDNREKIEKILTFRIPYYVGPLARGNSRFAWMTRKSEETITPWNFEEVVDKGASAQSFIERMTNFDKNLPNEKVLPKHSLLYEYFTVYNELTKVKYVTEGMRKPAFLSGEQKKAIVDLLFKTNRKVTVKQLKEDYFKKIECFDSVEISGVEDRFNASLGTYHDLLKIIKDKDFLDNEENEDILEDIVLTLTLFEDREMIEERLKTYAHLFDDKVMKQLKRRRYTGWGRLSRKLINGIRDKQSGKTILDFLKSDGFANRNFMQLIHDDSLTFKEDIQKAQVSGQGDSLHEHIANLAGSPAIKKGILQTVKVVDELVKVMGRHKPENIVIEMARENQTTQKGQKNSRERMKRIEEGIKELGSQILKEHPVENTQLQNEKLYLYYLQNGRDMYVDQELDINRLSDYDVDAIVPQSFLKDDSIDNKVLTRSDKNRGKSDNVPSEEVVKKMKNYWRQLLNAKLITQRKFDNLTKAERGGLSELDKAGFIKRQLVETRQITKHVAQILDSRMNTKYDENDKLIREVKVITLKSKLVSDFRKDFQFYKVREINNYHHAHDAYLNAVVGTALIKKYPKLESEFVYGDYKVYDVRKMIAKSEQEIGKATAKYFFYSNIMNFFKTEITLANGEIRKRPLIETNGETGEIVWDKGRDFATVRKVLSMPQVNIVKKTEVQTGGFSKESILPKRNSDKLIARKKDWDPKKYGGFDSPTVAYSVLVVAKVEKGKSKKLKSVKELLGITIMERSSFEKNPIDFLEAKGYKEVKKDLIIKLPKYSLFELENGRKRMLASAGELQKGNELALPSKYVNFLYLASHYEKLKGSPEDNEQKQLFVEQHKHYLDEIIEQISEFSKRVILADANLDKVLSAYNKHRDKPIREQAENIIHLFTLTNLGAPAAFKYFDTTIDRKRYTSTKEVLDATLIHQSITGLYETRIDLSQLGGD SRADypydvpdyaSGSpkkkrkvEASGSGRASPGIPGSTRNHDQEFDPPKVYPPVPAEKRKPIRVLSLFDGIATGLLVLKDLGIQVDRYIASEVCEDSITVGMVRHQGKIMYVGDVRSVTQKHIQEWGPFDLVIGGSPCNDLSIVNPARKGLYEGTGRLFFEFYRLLHDARPKEGDDRPFFWLFENVVAMGVSDKRDISRFLESNPVMIDAKEVSAAHRARYFWGNLPGMNRPLASTVNDKLELQECLEHGRIAKFSKVRTITTRSNSIKQGKDQHFPVFMNEKEDILWCTEMERVFGFPVHYTDVSNMSRLARQRLLGRSWSVPVIRHLFAPLKEYFACVSSGNSNANSRGPSFSSGLVPLSLRGSHMGPMEIYKTVSAWKRQPVRVLSLFRNIDKVLKSLGFLESGSGSGGGTLKYVEDVTNVVRRDVEKWGPFDLVYGSTQPLGSSCDRCPGWYMFQFHRILQYALPRQESQRPFFWIFMDNLLLTEDDQETTTRFLQTEAVTLQDVRGRDYQNAMRVWSNIPGLKSKHAPLTPKEEEYLQAQVRSRSKLDAPKVDLLVKNCLLPLREYFKYFSQNSLPLSRADpkkkrkvGSGatnfsllkqagdveenpgpGSGatnfsllkqagdveenpgpselikenmhmklymegtvdnhhfkctsegegkpyegtqtmrikvveggplpfafdilatsflygsktfinhtqgipdffkqsfpegftwervttyedggvltatqdtslqdgcliynvkirgvnftsngpvmqkktlgweaftetlypadgglegrndmalklvggshlianikttyrskkpaknlkmpgvyyvdyrlerikeannetyveqhevavarycdlpsklghkln *
[0275] SEQ ID NO: 4 (p92 (KRAB - dCas9 - XTEN16 - Dnmt3A - Dnmt3L - P2A - P2A - BFP): KRAB (жирный шрифт, из Gilbert et al., Cell, 2013, 2014), линкеры (подчеркивание), dCas9 (курсив), метка HA (строчные буквы), SV40 СЯЛ (строчные буквы, курсив), XTEN16 (последовательность из 16 аминокислот), Dnmt3A (жирный шрифт, курсив, из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), линкер из 27 аминокислот (подчеркивание, курсив, из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), Dnmt3L (подчеркивание, жирный шрифт, из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), последовательность расщепления пептидом P2A (строчные буквы, жирный шрифт), BFP (строчные буквы, подчеркивание))
DAKSLTAWSRTLVTFKDVFVDFTREEWKLLDTAQQIVYRNVMLENYKNLVSLGYQLTKPDVILRLEKGEEP GGSGGGS MDKKYSIGLAIGTNSVGWAVITDEYKVPSKKFKVLGNTDRHSIKKNLIGALLFDSGETAEATRLKRTARRRYTRRKNRICYLQEIFSNEMAKVDDSFFHRLEESFLVEEDKKHERHPIFGNIVDEVAYHEKYPTIYHLRKKLVDSTDKADLRLIYLALAHMIKFRGHFLIEGDLNPDNSDVDKLFIQLVQTYNQLFEENPINASGVDAKAILSARLSKSRRLENLIAQLPGEKKNGLFGNLIALSLGLTPNFKSNFDLAEDAKLQLSKDTYDDDLDNLLAQIGDQYADLFLAAKNLSDAILLSDILRVNTEITKAPLSASMIKRYDEHHQDLTLLKALVRQQLPEKYKEIFFDQSKNGYAGYIDGGASQEEFYKFIKPILEKMDGTEELLVKLNREDLLRKQRTFDNGSIPHQIHLGELHAILRRQEDFYPFLKDNREKIEKILTFRIPYYVGPLARGNSRFAWMTRKSEETITPWNFEEVVDKGASAQSFIERMTNFDKNLPNEKVLPKHSLLYEYFTVYNELTKVKYVTEGMRKPAFLSGEQKKAIVDLLFKTNRKVTVKQLKEDYFKKIECFDSVEISGVEDRFNASLGTYHDLLKIIKDKDFLDNEENEDILEDIVLTLTLFEDREMIEERLKTYAHLFDDKVMKQLKRRRYTGWGRLSRKLINGIRDKQSGKTILDFLKSDGFANRNFMQLIHDDSLTFKEDIQKAQVSGQGDSLHEHIANLAGSPAIKKGILQTVKVVDELVKVMGRHKPENIVIEMARENQTTQKGQKNSRERMKRIEEGIKELGSQILKEHPVENTQLQNEKLYLYYLQNGRDMYVDQELDINRLSDYDVDAIVPQSFLKDDSIDNKVLTRSDKNRGKSDNVPSEEVVKKMKNYWRQLLNAKLITQRKFDNLTKAERGGLSELDKAGFIKRQLVETRQITKHVAQILDSRMNTKYDENDKLIREVKVITLKSKLVSDFRKDFQFYKVREINNYHHAHDAYLNAVVGTALIKKYPKLESEFVYGDYKVYDVRKMIAKSEQEIGKATAKYFFYSNIMNFFKTEITLANGEIRKRPLIETNGETGEIVWDKGRDFATVRKVLSMPQVNIVKKTEVQTGGFSKESILPKRNSDKLIARKKDWDPKKYGGFDSPTVAYSVLVVAKVEKGKSKKLKSVKELLGITIMERSSFEKNPIDFLEAKGYKEVKKDLIIKLPKYSLFELENGRKRMLASAGELQKGNELALPSKYVNFLYLASHYEKLKGSPEDNEQKQLFVEQHKHYLDEIIEQISEFSKRVILADANLDKVLSAYNKHRDKPIREQAENIIHLFTLTNLGAPAAFKYFDTTIDRKRYTSTKEVLDATLIHQSITGLYETRIDLSQLGGD SRADypydvpdyaSGSpkkkrkvSPGSGSETPGTSESATPESNHDQEFDPPKVYPPVPAEKRKPIRVLSLFDGIATGLLVLKDLGIQVDRYIASEVCEDSITVGMVRHQGKIMYVGDVRSVTQKHIQEWGPFDLVIGGSPCNDLSIVNPARKGLYEGTGRLFFEFYRLLHDARPKEGDDRPFFWLFENVVAMGVSDKRDISRFLESNPVMIDAKEVSAAHRARYFWGNLPGMNRPLASTVNDKLELQECLEHGRIAKFSKVRTITTRSNSIKQGKDQHFPVFMNEKEDILWCTEMERVFGFPVHYTDVSNMSRLARQRLLGRSWSVPVIRHLFAPLKEYFACVSSGNSNANSRGPSFSSGLVPLSLRGSHMGPMEIYKTVSAWKRQPVRVLSLFRNIDKVLKSLGFLESGSGSGGGTLKYVEDVTNVVRRDVEKWGPFDLVYGSTQPLGSSCDRCPGWYMFQFHRILQYALPRQESQRPFFWIFMDNLLLTEDDQETTTRFLQTEAVTLQDVRGRDYQNAMRVWSNIPGLKSKHAPLTPKEEEYLQAQVRSRSKLDAPKVDLLVKNCLLPLREYFKYFSQNSLPLSRADpkkkrkvGSGatnfsllkqagdveenpgpGSGatnfsllkqagdveenpgpselikenmhmklymegtvdnhhfkctsegegkpyegtqtmrikvveggplpfafdilatsflygsktfinhtqgipdffkqsfpegftwervttyedggvltatqdtslqdgcliynvkirgvnftsngpvmqkktlgweaftetlypadgglegrndmalklvggshlianikttyrskkpaknlkmpgvyyvdyrlerikeannetyveqhevavarycdlpsklghkln *
[0276] SEQ ID NO: 5 (p93 (KRAB - dCas9 - XTEN80 - Dnmt3A - Dnmt3L - P2A - BFP): KRAB (жирный шрифт, из Gilbert et al., Cell, 2013, 2014), линкеры (подчеркивание), dCas9 (курсив), метка HA (строчные буквы), SV40 СЯЛ (строчные буквы, курсив), XTEN80 (последовательность из 80 аминокислот), Dnmt3A (жирный шрифт, курсив, из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), линкер из 27 аминокислот (подчеркивание, курсив; из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), Dnmt3L (подчеркивание, жирный шрифт, из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), последовательность расщепления пептидом P2A (строчные буквы, жирный шрифт), BFP (строчные буквы, подчеркивание))
DAKSLTAWSRTLVTFKDVFVDFTREEWKLLDTAQQIVYRNVMLENYKNLVSLGYQLTKPDVILRLEKGEEP GGSGGGS MDKKYSIGLAIGTNSVGWAVITDEYKVPSKKFKVLGNTDRHSIKKNLIGALLFDSGETAEATRLKRTARRRYTRRKNRICYLQEIFSNEMAKVDDSFFHRLEESFLVEEDKKHERHPIFGNIVDEVAYHEKYPTIYHLRKKLVDSTDKADLRLIYLALAHMIKFRGHFLIEGDLNPDNSDVDKLFIQLVQTYNQLFEENPINASGVDAKAILSARLSKSRRLENLIAQLPGEKKNGLFGNLIALSLGLTPNFKSNFDLAEDAKLQLSKDTYDDDLDNLLAQIGDQYADLFLAAKNLSDAILLSDILRVNTEITKAPLSASMIKRYDEHHQDLTLLKALVRQQLPEKYKEIFFDQSKNGYAGYIDGGASQEEFYKFIKPILEKMDGTEELLVKLNREDLLRKQRTFDNGSIPHQIHLGELHAILRRQEDFYPFLKDNREKIEKILTFRIPYYVGPLARGNSRFAWMTRKSEETITPWNFEEVVDKGASAQSFIERMTNFDKNLPNEKVLPKHSLLYEYFTVYNELTKVKYVTEGMRKPAFLSGEQKKAIVDLLFKTNRKVTVKQLKEDYFKKIECFDSVEISGVEDRFNASLGTYHDLLKIIKDKDFLDNEENEDILEDIVLTLTLFEDREMIEERLKTYAHLFDDKVMKQLKRRRYTGWGRLSRKLINGIRDKQSGKTILDFLKSDGFANRNFMQLIHDDSLTFKEDIQKAQVSGQGDSLHEHIANLAGSPAIKKGILQTVKVVDELVKVMGRHKPENIVIEMARENQTTQKGQKNSRERMKRIEEGIKELGSQILKEHPVENTQLQNEKLYLYYLQNGRDMYVDQELDINRLSDYDVDAIVPQSFLKDDSIDNKVLTRSDKNRGKSDNVPSEEVVKKMKNYWRQLLNAKLITQRKFDNLTKAERGGLSELDKAGFIKRQLVETRQITKHVAQILDSRMNTKYDENDKLIREVKVITLKSKLVSDFRKDFQFYKVREINNYHHAHDAYLNAVVGTALIKKYPKLESEFVYGDYKVYDVRKMIAKSEQEIGKATAKYFFYSNIMNFFKTEITLANGEIRKRPLIETNGETGEIVWDKGRDFATVRKVLSMPQVNIVKKTEVQTGGFSKESILPKRNSDKLIARKKDWDPKKYGGFDSPTVAYSVLVVAKVEKGKSKKLKSVKELLGITIMERSSFEKNPIDFLEAKGYKEVKKDLIIKLPKYSLFELENGRKRMLASAGELQKGNELALPSKYVNFLYLASHYEKLKGSPEDNEQKQLFVEQHKHYLDEIIEQISEFSKRVILADANLDKVLSAYNKHRDKPIREQAENIIHLFTLTNLGAPAAFKYFDTTIDRKRYTSTKEVLDATLIHQSITGLYETRIDLSQLGGD SRADypydvpdyaSGSpkkkrkvSPGGGPSSGAPPPSGGSPAGSPTSTEEGTSESATPESGPGTSTEPSEGSAPGSPAGSPTSTEEGTSTEPSEGSAPGTSTEPSENHDQEFDPPKVYPPVPAEKRKPIRVLSLFDGIATGLLVLKDLGIQVDRYIASEVCEDSITVGMVRHQGKIMYVGDVRSVTQKHIQEWGPFDLVIGGSPCNDLSIVNPARKGLYEGTGRLFFEFYRLLHDARPKEGDDRPFFWLFENVVAMGVSDKRDISRFLESNPVMIDAKEVSAAHRARYFWGNLPGMNRPLASTVNDKLELQECLEHGRIAKFSKVRTITTRSNSIKQGKDQHFPVFMNEKEDILWCTEMERVFGFPVHYTDVSNMSRLARQRLLGRSWSVPVIRHLFAPLKEYFACVSSGNSNANSRGPSFSSGLVPLSLRGSHMGPMEIYKTVSAWKRQPVRVLSLFRNIDKVLKSLGFLESGSGSGGGTLKYVEDVTNVVRRDVEKWGPFDLVYGSTQPLGSSCDRCPGWYMFQFHRILQYALPRQESQRPFFWIFMDNLLLTEDDQETTTRFLQTEAVTLQDVRGRDYQNAMRVWSNIPGLKSKHAPLTPKEEEYLQAQVRSRSKLDAPKVDLLVKNCLLPLREYFKYFSQNSLPLSRADpkkkrkvGSGatnfsllkqagdveenpgpselikenmhmklymegtvdnhhfkctsegegkpyegtqtmrikvveggplpfafdilatsflygsktfinhtqgipdffkqsfpegftwervttyedggvltatqdtslqdgcliynvkirgvnftsngpvmqkktlgweaftetlypadgglegrndmalklvggshlianikttyrskkpaknlkmpgvyyvdyrlerikeannetyveqhevavarycdlpsklghkln *
[0277] SEQ ID NO: 6 (p94 (KRAB - dCas9 - XTEN80 - Dnmt3A - Dnmt3L - P2A - P2A - BFP): KRAB (жирный шрифт, из Gilbert et al., Cell, 2013, 2014), линкеры (подчеркивание), dCas9 (курсив), метка HA (строчные буквы), SV40 СЯЛ (строчные буквы, курсив), XTEN80 (последовательность из 80 аминокислот), Dnmt3A (жирный шрифт, курсив, из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), линкер из 27 аминокислот (подчеркивание, курсив, из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), Dnmt3L (подчеркивание, жирный шрифт, из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), последовательность расщепления пептидом P2A (строчные буквы, жирный шрифт), BFP (строчные буквы, подчеркивание))
DAKSLTAWSRTLVTFKDVFVDFTREEWKLLDTAQQIVYRNVMLENYKNLVSLGYQLTKPDVILRLEKGEEP GGSGGGS MDKKYSIGLAIGTNSVGWAVITDEYKVPSKKFKVLGNTDRHSIKKNLIGALLFDSGETAEATRLKRTARRRYTRRKNRICYLQEIFSNEMAKVDDSFFHRLEESFLVEEDKKHERHPIFGNIVDEVAYHEKYPTIYHLRKKLVDSTDKADLRLIYLALAHMIKFRGHFLIEGDLNPDNSDVDKLFIQLVQTYNQLFEENPINASGVDAKAILSARLSKSRRLENLIAQLPGEKKNGLFGNLIALSLGLTPNFKSNFDLAEDAKLQLSKDTYDDDLDNLLAQIGDQYADLFLAAKNLSDAILLSDILRVNTEITKAPLSASMIKRYDEHHQDLTLLKALVRQQLPEKYKEIFFDQSKNGYAGYIDGGASQEEFYKFIKPILEKMDGTEELLVKLNREDLLRKQRTFDNGSIPHQIHLGELHAILRRQEDFYPFLKDNREKIEKILTFRIPYYVGPLARGNSRFAWMTRKSEETITPWNFEEVVDKGASAQSFIERMTNFDKNLPNEKVLPKHSLLYEYFTVYNELTKVKYVTEGMRKPAFLSGEQKKAIVDLLFKTNRKVTVKQLKEDYFKKIECFDSVEISGVEDRFNASLGTYHDLLKIIKDKDFLDNEENEDILEDIVLTLTLFEDREMIEERLKTYAHLFDDKVMKQLKRRRYTGWGRLSRKLINGIRDKQSGKTILDFLKSDGFANRNFMQLIHDDSLTFKEDIQKAQVSGQGDSLHEHIANLAGSPAIKKGILQTVKVVDELVKVMGRHKPENIVIEMARENQTTQKGQKNSRERMKRIEEGIKELGSQILKEHPVENTQLQNEKLYLYYLQNGRDMYVDQELDINRLSDYDVDAIVPQSFLKDDSIDNKVLTRSDKNRGKSDNVPSEEVVKKMKNYWRQLLNAKLITQRKFDNLTKAERGGLSELDKAGFIKRQLVETRQITKHVAQILDSRMNTKYDENDKLIREVKVITLKSKLVSDFRKDFQFYKVREINNYHHAHDAYLNAVVGTALIKKYPKLESEFVYGDYKVYDVRKMIAKSEQEIGKATAKYFFYSNIMNFFKTEITLANGEIRKRPLIETNGETGEIVWDKGRDFATVRKVLSMPQVNIVKKTEVQTGGFSKESILPKRNSDKLIARKKDWDPKKYGGFDSPTVAYSVLVVAKVEKGKSKKLKSVKELLGITIMERSSFEKNPIDFLEAKGYKEVKKDLIIKLPKYSLFELENGRKRMLASAGELQKGNELALPSKYVNFLYLASHYEKLKGSPEDNEQKQLFVEQHKHYLDEIIEQISEFSKRVILADANLDKVLSAYNKHRDKPIREQAENIIHLFTLTNLGAPAAFKYFDTTIDRKRYTSTKEVLDATLIHQSITGLYETRIDLSQLGGD SRADypydvpdyaSGSpkkkrkvSPGGGPSSGAPPPSGGSPAGSPTSTEEGTSESATPESGPGTSTEPSEGSAPGSPAGSPTSTEEGTSTEPSEGSAPGTSTEPSENHDQEFDPPKVYPPVPAEKRKPIRVLSLFDGIATGLLVLKDLGIQVDRYIASEVCEDSITVGMVRHQGKIMYVGDVRSVTQKHIQEWGPFDLVIGGSPCNDLSIVNPARKGLYEGTGRLFFEFYRLLHDARPKEGDDRPFFWLFENVVAMGVSDKRDISRFLESNPVMIDAKEVSAAHRARYFWGNLPGMNRPLASTVNDKLELQECLEHGRIAKFSKVRTITTRSNSIKQGKDQHFPVFMNEKEDILWCTEMERVFGFPVHYTDVSNMSRLARQRLLGRSWSVPVIRHLFAPLKEYFACVSSGNSNANSRGPSFSSGLVPLSLRGSHMGPMEIYKTVSAWKRQPVRVLSLFRNIDKVLKSLGFLESGSGSGGGTLKYVEDVTNVVRRDVEKWGPFDLVYGSTQPLGSSCDRCPGWYMFQFHRILQYALPRQESQRPFFWIFMDNLLLTEDDQETTTRFLQTEAVTLQDVRGRDYQNAMRVWSNIPGLKSKHAPLTPKEEEYLQAQVRSRSKLDAPKVDLLVKNCLLPLREYFKYFSQNSLPLSRADpkkkrkvGSGatnfsllkqagdveenpgpGSGatnfsllkqagdveenpgpselikenmhmklymegtvdnhhfkctsegegkpyegtqtmrikvveggplpfafdilatsflygsktfinhtqgipdffkqsfpegftwervttyedggvltatqdtslqdgcliynvkirgvnftsngpvmqkktlgweaftetlypadgglegrndmalklvggshlianikttyrskkpaknlkmpgvyyvdyrlerikeannetyveqhevavarycdlpsklghkln *
[0278] SEQ ID NO: 7 (p95 (KRAB - XTEN16 - dCas9 - Dnmt3A - Dnmt3L - P2A - BFP): KRAB (жирный шрифт, из Gilbert et al., Cell, 2013, 2014), линкеры (подчеркивание), XTEN16 (последовательность из 16 аминокислот), dCas9 (курсив), метка HA (строчные буквы), SV40 СЯЛ (строчные буквы, курсив), Dnmt3A (жирный шрифт, курсив, из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), линкер из 27 аминокислот (подчеркивание, курсив, из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), Dnmt3L (подчеркивание, жирный шрифт, из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), последовательность расщепления пептидом P2A (строчные буквы, жирный шрифт), BFP (строчные буквы, подчеркивание))
DAKSLTAWSRTLVTFKDVFVDFTREEWKLLDTAQQIVYRNVMLENYKNLVSLGYQLTKPDVILRLEKGEEPSGSETPGTSESATPESMDKKYSIGLAIGTNSVGWAVITDEYKVPSKKFKVLGNTDRHSIKKNLIGALLFDSGETAEATRLKRTARRRYTRRKNRICYLQEIFSNEMAKVDDSFFHRLEESFLVEEDKKHERHPIFGNIVDEVAYHEKYPTIYHLRKKLVDSTDKADLRLIYLALAHMIKFRGHFLIEGDLNPDNSDVDKLFIQLVQTYNQLFEENPINASGVDAKAILSARLSKSRRLENLIAQLPGEKKNGLFGNLIALSLGLTPNFKSNFDLAEDAKLQLSKDTYDDDLDNLLAQIGDQYADLFLAAKNLSDAILLSDILRVNTEITKAPLSASMIKRYDEHHQDLTLLKALVRQQLPEKYKEIFFDQSKNGYAGYIDGGASQEEFYKFIKPILEKMDGTEELLVKLNREDLLRKQRTFDNGSIPHQIHLGELHAILRRQEDFYPFLKDNREKIEKILTFRIPYYVGPLARGNSRFAWMTRKSEETITPWNFEEVVDKGASAQSFIERMTNFDKNLPNEKVLPKHSLLYEYFTVYNELTKVKYVTEGMRKPAFLSGEQKKAIVDLLFKTNRKVTVKQLKEDYFKKIECFDSVEISGVEDRFNASLGTYHDLLKIIKDKDFLDNEENEDILEDIVLTLTLFEDREMIEERLKTYAHLFDDKVMKQLKRRRYTGWGRLSRKLINGIRDKQSGKTILDFLKSDGFANRNFMQLIHDDSLTFKEDIQKAQVSGQGDSLHEHIANLAGSPAIKKGILQTVKVVDELVKVMGRHKPENIVIEMARENQTTQKGQKNSRERMKRIEEGIKELGSQILKEHPVENTQLQNEKLYLYYLQNGRDMYVDQELDINRLSDYDVDAIVPQSFLKDDSIDNKVLTRSDKNRGKSDNVPSEEVVKKMKNYWRQLLNAKLITQRKFDNLTKAERGGLSELDKAGFIKRQLVETRQITKHVAQILDSRMNTKYDENDKLIREVKVITLKSKLVSDFRKDFQFYKVREINNYHHAHDAYLNAVVGTALIKKYPKLESEFVYGDYKVYDVRKMIAKSEQEIGKATAKYFFYSNIMNFFKTEITLANGEIRKRPLIETNGETGEIVWDKGRDFATVRKVLSMPQVNIVKKTEVQTGGFSKESILPKRNSDKLIARKKDWDPKKYGGFDSPTVAYSVLVVAKVEKGKSKKLKSVKELLGITIMERSSFEKNPIDFLEAKGYKEVKKDLIIKLPKYSLFELENGRKRMLASAGELQKGNELALPSKYVNFLYLASHYEKLKGSPEDNEQKQLFVEQHKHYLDEIIEQISEFSKRVILADANLDKVLSAYNKHRDKPIREQAENIIHLFTLTNLGAPAAFKYFDTTIDRKRYTSTKEVLDATLIHQSITGLYETRIDLSQLGGDSRADypydvpdyaSGSpkkkrkvEASGSGRASPGIPGSTRNHDQEFDPPKVYPPVPAEKRKPIRVLSLFDGIATGLLVLKDLGIQVDRYIASEVCEDSITVGMVRHQGKIMYVGDVRSVTQKHIQEWGPFDLVIGGSPCNDLSIVNPARKGLYEGTGRLFFEFYRLLHDARPKEGDDRPFFWLFENVVAMGVSDKRDISRFLESNPVMIDAKEVSAAHRARYFWGNLPGMNRPLASTVNDKLELQECLEHGRIAKFSKVRTITTRSNSIKQGKDQHFPVFMNEKEDILWCTEMERVFGFPVHYTDVSNMSRLARQRLLGRSWSVPVIRHLFAPLKEYFACVSSGNSNANSRGPSFSSGLVPLSLRGSHMGPMEIYKTVSAWKRQPVRVLSLFRNIDKVLKSLGFLESGSGSGGGTLKYVEDVTNVVRRDVEKWGPFDLVYGSTQPLGSSCDRCPGWYMFQFHRILQYALPRQESQRPFFWIFMDNLLLTEDDQETTTRFLQTEAVTLQDVRGRDYQNAMRVWSNIPGLKSKHAPLTPKEEEYLQAQVRSRSKLDAPKVDLLVKNCLLPLREYFKYFSQNSLPLSRADpkkkrkvGSGatnfsllkqagdveenpgpselikenmhmklymegtvdnhhfkctsegegkpyegtqtmrikvveggplpfafdilatsflygsktfinhtqgipdffkqsfpegftwervttyedggvltatqdtslqdgcliynvkirgvnftsngpvmqkktlgweaftetlypadgglegrndmalklvggshlianikttyrskkpaknlkmpgvyyvdyrlerikeannetyveqhevavarycdlpsklghkln *
[0279] SEQ ID NO: 8 (p96 (KRAB - XTEN16 - dCas9 - Dnmt3A - Dnmt3L - P2A - P2A - BFP): KRAB (жирный шрифт, из Gilbert et al., Cell, 2013, 2014), линкеры (подчеркивание), XTEN16 (последовательность из 16 аминокислот), dCas9 (курсив), метка HA (строчные буквы), SV40 СЯЛ (строчные буквы, курсив), Dnmt3A (жирный шрифт, курсив, из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), линкер из 27 аминокислот (подчеркивание, курсив, из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), Dnmt3L (подчеркивание, жирный шрифт, из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), последовательность расщепления пептидом P2A (строчные буквы, жирный шрифт); BFP (строчные буквы, подчеркивание))
DAKSLTAWSRTLVTFKDVFVDFTREEWKLLDTAQQIVYRNVMLENYKNLVSLGYQLTKPDVILRLEKGEEPSGSETPGTSESATPESMDKKYSIGLAIGTNSVGWAVITDEYKVPSKKFKVLGNTDRHSIKKNLIGALLFDSGETAEATRLKRTARRRYTRRKNRICYLQEIFSNEMAKVDDSFFHRLEESFLVEEDKKHERHPIFGNIVDEVAYHEKYPTIYHLRKKLVDSTDKADLRLIYLALAHMIKFRGHFLIEGDLNPDNSDVDKLFIQLVQTYNQLFEENPINASGVDAKAILSARLSKSRRLENLIAQLPGEKKNGLFGNLIALSLGLTPNFKSNFDLAEDAKLQLSKDTYDDDLDNLLAQIGDQYADLFLAAKNLSDAILLSDILRVNTEITKAPLSASMIKRYDEHHQDLTLLKALVRQQLPEKYKEIFFDQSKNGYAGYIDGGASQEEFYKFIKPILEKMDGTEELLVKLNREDLLRKQRTFDNGSIPHQIHLGELHAILRRQEDFYPFLKDNREKIEKILTFRIPYYVGPLARGNSRFAWMTRKSEETITPWNFEEVVDKGASAQSFIERMTNFDKNLPNEKVLPKHSLLYEYFTVYNELTKVKYVTEGMRKPAFLSGEQKKAIVDLLFKTNRKVTVKQLKEDYFKKIECFDSVEISGVEDRFNASLGTYHDLLKIIKDKDFLDNEENEDILEDIVLTLTLFEDREMIEERLKTYAHLFDDKVMKQLKRRRYTGWGRLSRKLINGIRDKQSGKTILDFLKSDGFANRNFMQLIHDDSLTFKEDIQKAQVSGQGDSLHEHIANLAGSPAIKKGILQTVKVVDELVKVMGRHKPENIVIEMARENQTTQKGQKNSRERMKRIEEGIKELGSQILKEHPVENTQLQNEKLYLYYLQNGRDMYVDQELDINRLSDYDVDAIVPQSFLKDDSIDNKVLTRSDKNRGKSDNVPSEEVVKKMKNYWRQLLNAKLITQRKFDNLTKAERGGLSELDKAGFIKRQLVETRQITKHVAQILDSRMNTKYDENDKLIREVKVITLKSKLVSDFRKDFQFYKVREINNYHHAHDAYLNAVVGTALIKKYPKLESEFVYGDYKVYDVRKMIAKSEQEIGKATAKYFFYSNIMNFFKTEITLANGEIRKRPLIETNGETGEIVWDKGRDFATVRKVLSMPQVNIVKKTEVQTGGFSKESILPKRNSDKLIARKKDWDPKKYGGFDSPTVAYSVLVVAKVEKGKSKKLKSVKELLGITIMERSSFEKNPIDFLEAKGYKEVKKDLIIKLPKYSLFELENGRKRMLASAGELQKGNELALPSKYVNFLYLASHYEKLKGSPEDNEQKQLFVEQHKHYLDEIIEQISEFSKRVILADANLDKVLSAYNKHRDKPIREQAENIIHLFTLTNLGAPAAFKYFDTTIDRKRYTSTKEVLDATLIHQSITGLYETRIDLSQLGGDSRADypydvpdyaSGSpkkkrkvEASGSGRASPGIPGSTRNHDQEFDPPKVYPPVPAEKRKPIRVLSLFDGIATGLLVLKDLGIQVDRYIASEVCEDSITVGMVRHQGKIMYVGDVRSVTQKHIQEWGPFDLVIGGSPCNDLSIVNPARKGLYEGTGRLFFEFYRLLHDARPKEGDDRPFFWLFENVVAMGVSDKRDISRFLESNPVMIDAKEVSAAHRARYFWGNLPGMNRPLASTVNDKLELQECLEHGRIAKFSKVRTITTRSNSIKQGKDQHFPVFMNEKEDILWCTEMERVFGFPVHYTDVSNMSRLARQRLLGRSWSVPVIRHLFAPLKEYFACVSSGNSNANSRGPSFSSGLVPLSLRGSHMGPMEIYKTVSAWKRQPVRVLSLFRNIDKVLKSLGFLESGSGSGGGTLKYVEDVTNVVRRDVEKWGPFDLVYGSTQPLGSSCDRCPGWYMFQFHRILQYALPRQESQRPFFWIFMDNLLLTEDDQETTTRFLQTEAVTLQDVRGRDYQNAMRVWSNIPGLKSKHAPLTPKEEEYLQAQVRSRSKLDAPKVDLLVKNCLLPLREYFKYFSQNSLPLSRADpkkkrkvGSGatnfsllkqagdveenpgpGSGatnfsllkqagdveenpgpselikenmhmklymegtvdnhhfkctsegegkpyegtqtmrikvveggplpfafdilatsflygsktfinhtqgipdffkqsfpegftwervttyedggvltatqdtslqdgcliynvkirgvnftsngpvmqkktlgweaftetlypadgglegrndmalklvggshlianikttyrskkpaknlkmpgvyyvdyrlerikeannetyveqhevavarycdlpsklghkln *
[0280] SEQ ID NO: 9 (p97 (KRAB - XTEN80 - dCas9 - Dnmt3A - Dnmt3L - P2A - BFP): KRAB (жирный шрифт, из Gilbert et al., Cell, 2013, 2014), линкеры (подчеркивание), XTEN80 (последовательность из 80 аминокислот), dCas9 (курсив); метка HA (строчные буквы), SV40 СЯЛ (строчные буквы, курсив), Dnmt3A (жирный шрифт, курсив, из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), линкер из 27 аминокислот (подчеркивание, курсив, из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), Dnmt3L (подчеркивание, жирный шрифт, из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), последовательность расщепления пептидом P2A (строчные буквы, жирный шрифт), BFP (строчные буквы, подчеркивание))
DAKSLTAWSRTLVTFKDVFVDFTREEWKLLDTAQQIVYRNVMLENYKNLVSLGYQLTKPDVILRLEKGEEPGGPSSGAPPPSGGSPAGSPTSTEEGTSESATPESGPGTSTEPSEGSAPGSPAGSPTSTEEGTSTEPSEGSAPGTSTEPSEMDKKYSIGLAIGTNSVGWAVITDEYKVPSKKFKVLGNTDRHSIKKNLIGALLFDSGETAEATRLKRTARRRYTRRKNRICYLQEIFSNEMAKVDDSFFHRLEESFLVEEDKKHERHPIFGNIVDEVAYHEKYPTIYHLRKKLVDSTDKADLRLIYLALAHMIKFRGHFLIEGDLNPDNSDVDKLFIQLVQTYNQLFEENPINASGVDAKAILSARLSKSRRLENLIAQLPGEKKNGLFGNLIALSLGLTPNFKSNFDLAEDAKLQLSKDTYDDDLDNLLAQIGDQYADLFLAAKNLSDAILLSDILRVNTEITKAPLSASMIKRYDEHHQDLTLLKALVRQQLPEKYKEIFFDQSKNGYAGYIDGGASQEEFYKFIKPILEKMDGTEELLVKLNREDLLRKQRTFDNGSIPHQIHLGELHAILRRQEDFYPFLKDNREKIEKILTFRIPYYVGPLARGNSRFAWMTRKSEETITPWNFEEVVDKGASAQSFIERMTNFDKNLPNEKVLPKHSLLYEYFTVYNELTKVKYVTEGMRKPAFLSGEQKKAIVDLLFKTNRKVTVKQLKEDYFKKIECFDSVEISGVEDRFNASLGTYHDLLKIIKDKDFLDNEENEDILEDIVLTLTLFEDREMIEERLKTYAHLFDDKVMKQLKRRRYTGWGRLSRKLINGIRDKQSGKTILDFLKSDGFANRNFMQLIHDDSLTFKEDIQKAQVSGQGDSLHEHIANLAGSPAIKKGILQTVKVVDELVKVMGRHKPENIVIEMARENQTTQKGQKNSRERMKRIEEGIKELGSQILKEHPVENTQLQNEKLYLYYLQNGRDMYVDQELDINRLSDYDVDAIVPQSFLKDDSIDNKVLTRSDKNRGKSDNVPSEEVVKKMKNYWRQLLNAKLITQRKFDNLTKAERGGLSELDKAGFIKRQLVETRQITKHVAQILDSRMNTKYDENDKLIREVKVITLKSKLVSDFRKDFQFYKVREINNYHHAHDAYLNAVVGTALIKKYPKLESEFVYGDYKVYDVRKMIAKSEQEIGKATAKYFFYSNIMNFFKTEITLANGEIRKRPLIETNGETGEIVWDKGRDFATVRKVLSMPQVNIVKKTEVQTGGFSKESILPKRNSDKLIARKKDWDPKKYGGFDSPTVAYSVLVVAKVEKGKSKKLKSVKELLGITIMERSSFEKNPIDFLEAKGYKEVKKDLIIKLPKYSLFELENGRKRMLASAGELQKGNELALPSKYVNFLYLASHYEKLKGSPEDNEQKQLFVEQHKHYLDEIIEQISEFSKRVILADANLDKVLSAYNKHRDKPIREQAENIIHLFTLTNLGAPAAFKYFDTTIDRKRYTSTKEVLDATLIHQSITGLYETRIDLSQLGGDSRADypydvpdyaSGSpkkkrkvEASGSGRASPGIPGSTRNHDQEFDPPKVYPPVPAEKRKPIRVLSLFDGIATGLLVLKDLGIQVDRYIASEVCEDSITVGMVRHQGKIMYVGDVRSVTQKHIQEWGPFDLVIGGSPCNDLSIVNPARKGLYEGTGRLFFEFYRLLHDARPKEGDDRPFFWLFENVVAMGVSDKRDISRFLESNPVMIDAKEVSAAHRARYFWGNLPGMNRPLASTVNDKLELQECLEHGRIAKFSKVRTITTRSNSIKQGKDQHFPVFMNEKEDILWCTEMERVFGFPVHYTDVSNMSRLARQRLLGRSWSVPVIRHLFAPLKEYFACVSSGNSNANSRGPSFSSGLVPLSLRGSHMGPMEIYKTVSAWKRQPVRVLSLFRNIDKVLKSLGFLESGSGSGGGTLKYVEDVTNVVRRDVEKWGPFDLVYGSTQPLGSSCDRCPGWYMFQFHRILQYALPRQESQRPFFWIFMDNLLLTEDDQETTTRFLQTEAVTLQDVRGRDYQNAMRVWSNIPGLKSKHAPLTPKEEEYLQAQVRSRSKLDAPKVDLLVKNCLLPLREYFKYFSQNSLPLSRADpkkkrkvGSGatnfsllkqagdveenpgpselikenmhmklymegtvdnhhfkctsegegkpyegtqtmrikvveggplpfafdilatsflygsktfinhtqgipdffkqsfpegftwervttyedggvltatqdtslqdgcliynvkirgvnftsngpvmqkktlgweaftetlypadgglegrndmalklvggshlianikttyrskkpaknlkmpgvyyvdyrlerikeannetyveqhevavarycdlpsklghkln *
[0281] SEQ ID NO: 10 (p98 (KRAB - XTEN80 - dCas9 - Dnmt3A - Dnmt3L - P2A - P2A - BFP): KRAB (жирный шрифт, из Gilbert et al., Cell, 2013, 2014), линкеры (подчеркивание), XTEN80 (последовательность из 80 аминокислот), dCas9 (курсив), метка HA (строчные буквы), SV40 СЯЛ (строчные буквы, курсив), Dnmt3A (жирный шрифт, курсив, из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), линкер из 27 аминокислот (подчеркивание, курсив, из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), Dnmt3L (подчеркивание, жирный шрифт, из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), последовательность расщепления пептидом P2A (строчные буквы, жирный шрифт), BFP (строчные буквы, подчеркивание))
DAKSLTAWSRTLVTFKDVFVDFTREEWKLLDTAQQIVYRNVMLENYKNLVSLGYQLTKPDVILRLEKGEEPGGPSSGAPPPSGGSPAGSPTSTEEGTSESATPESGPGTSTEPSEGSAPGSPAGSPTSTEEGTSTEPSEGSAPGTSTEPSEMDKKYSIGLAIGTNSVGWAVITDEYKVPSKKFKVLGNTDRHSIKKNLIGALLFDSGETAEATRLKRTARRRYTRRKNRICYLQEIFSNEMAKVDDSFFHRLEESFLVEEDKKHERHPIFGNIVDEVAYHEKYPTIYHLRKKLVDSTDKADLRLIYLALAHMIKFRGHFLIEGDLNPDNSDVDKLFIQLVQTYNQLFEENPINASGVDAKAILSARLSKSRRLENLIAQLPGEKKNGLFGNLIALSLGLTPNFKSNFDLAEDAKLQLSKDTYDDDLDNLLAQIGDQYADLFLAAKNLSDAILLSDILRVNTEITKAPLSASMIKRYDEHHQDLTLLKALVRQQLPEKYKEIFFDQSKNGYAGYIDGGASQEEFYKFIKPILEKMDGTEELLVKLNREDLLRKQRTFDNGSIPHQIHLGELHAILRRQEDFYPFLKDNREKIEKILTFRIPYYVGPLARGNSRFAWMTRKSEETITPWNFEEVVDKGASAQSFIERMTNFDKNLPNEKVLPKHSLLYEYFTVYNELTKVKYVTEGMRKPAFLSGEQKKAIVDLLFKTNRKVTVKQLKEDYFKKIECFDSVEISGVEDRFNASLGTYHDLLKIIKDKDFLDNEENEDILEDIVLTLTLFEDREMIEERLKTYAHLFDDKVMKQLKRRRYTGWGRLSRKLINGIRDKQSGKTILDFLKSDGFANRNFMQLIHDDSLTFKEDIQKAQVSGQGDSLHEHIANLAGSPAIKKGILQTVKVVDELVKVMGRHKPENIVIEMARENQTTQKGQKNSRERMKRIEEGIKELGSQILKEHPVENTQLQNEKLYLYYLQNGRDMYVDQELDINRLSDYDVDAIVPQSFLKDDSIDNKVLTRSDKNRGKSDNVPSEEVVKKMKNYWRQLLNAKLITQRKFDNLTKAERGGLSELDKAGFIKRQLVETRQITKHVAQILDSRMNTKYDENDKLIREVKVITLKSKLVSDFRKDFQFYKVREINNYHHAHDAYLNAVVGTALIKKYPKLESEFVYGDYKVYDVRKMIAKSEQEIGKATAKYFFYSNIMNFFKTEITLANGEIRKRPLIETNGETGEIVWDKGRDFATVRKVLSMPQVNIVKKTEVQTGGFSKESILPKRNSDKLIARKKDWDPKKYGGFDSPTVAYSVLVVAKVEKGKSKKLKSVKELLGITIMERSSFEKNPIDFLEAKGYKEVKKDLIIKLPKYSLFELENGRKRMLASAGELQKGNELALPSKYVNFLYLASHYEKLKGSPEDNEQKQLFVEQHKHYLDEIIEQISEFSKRVILADANLDKVLSAYNKHRDKPIREQAENIIHLFTLTNLGAPAAFKYFDTTIDRKRYTSTKEVLDATLIHQSITGLYETRIDLSQLGGDSRADypydvpdyaSGSpkkkrkvEASGSGRASPGIPGSTRNHDQEFDPPKVYPPVPAEKRKPIRVLSLFDGIATGLLVLKDLGIQVDRYIASEVCEDSITVGMVRHQGKIMYVGDVRSVTQKHIQEWGPFDLVIGGSPCNDLSIVNPARKGLYEGTGRLFFEFYRLLHDARPKEGDDRPFFWLFENVVAMGVSDKRDISRFLESNPVMIDAKEVSAAHRARYFWGNLPGMNRPLASTVNDKLELQECLEHGRIAKFSKVRTITTRSNSIKQGKDQHFPVFMNEKEDILWCTEMERVFGFPVHYTDVSNMSRLARQRLLGRSWSVPVIRHLFAPLKEYFACVSSGNSNANSRGPSFSSGLVPLSLRGSHMGPMEIYKTVSAWKRQPVRVLSLFRNIDKVLKSLGFLESGSGSGGGTLKYVEDVTNVVRRDVEKWGPFDLVYGSTQPLGSSCDRCPGWYMFQFHRILQYALPRQESQRPFFWIFMDNLLLTEDDQETTTRFLQTEAVTLQDVRGRDYQNAMRVWSNIPGLKSKHAPLTPKEEEYLQAQVRSRSKLDAPKVDLLVKNCLLPLREYFKYFSQNSLPLSRADpkkkrkvGSGatnfsllkqagdveenpgpGSGatnfsllkqagdveenpgpselikenmhmklymegtvdnhhfkctsegegkpyegtqtmrikvveggplpfafdilatsflygsktfinhtqgipdffkqsfpegftwervttyedggvltatqdtslqdgcliynvkirgvnftsngpvmqkktlgweaftetlypadgglegrndmalklvggshlianikttyrskkpaknlkmpgvyyvdyrlerikeannetyveqhevavarycdlpsklghkln *
[0282] SEQ ID NO: 11 (p99 (KRAB - XTEN16 - dCas9 - XTEN80 - Dnmt3A - Dnmt3L - P2A - BFP): KRAB (жирный шрифт, из Gilbert et al., Cell, 2013, 2014), XTEN16 (последовательность из 16 аминокислот), dCas9 (курсив), метка HA (строчные буквы), линкеры (подчеркивание), SV40 СЯЛ (строчные буквы, курсив), XTEN80 (строчные буквы, жирный шрифт, курсив, последовательность из 80 аминокислот), Dnmt3A (жирный шрифт, курсив, из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), линкер из 27 аминокислот (подчеркивание, курсив, из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), Dnmt3L (подчеркивание, жирный шрифт, из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), последовательность расщепления пептидом P2A (строчные буквы, жирный шрифт), BFP (строчные буквы, подчеркивание))
DAKSLTAWSRTLVTFKDVFVDFTREEWKLLDTAQQIVYRNVMLENYKNLVSLGYQLTKPDVILRLEKGEEPSGSETPGTSESATPESMDKKYSIGLAIGTNSVGWAVITDEYKVPSKKFKVLGNTDRHSIKKNLIGALLFDSGETAEATRLKRTARRRYTRRKNRICYLQEIFSNEMAKVDDSFFHRLEESFLVEEDKKHERHPIFGNIVDEVAYHEKYPTIYHLRKKLVDSTDKADLRLIYLALAHMIKFRGHFLIEGDLNPDNSDVDKLFIQLVQTYNQLFEENPINASGVDAKAILSARLSKSRRLENLIAQLPGEKKNGLFGNLIALSLGLTPNFKSNFDLAEDAKLQLSKDTYDDDLDNLLAQIGDQYADLFLAAKNLSDAILLSDILRVNTEITKAPLSASMIKRYDEHHQDLTLLKALVRQQLPEKYKEIFFDQSKNGYAGYIDGGASQEEFYKFIKPILEKMDGTEELLVKLNREDLLRKQRTFDNGSIPHQIHLGELHAILRRQEDFYPFLKDNREKIEKILTFRIPYYVGPLARGNSRFAWMTRKSEETITPWNFEEVVDKGASAQSFIERMTNFDKNLPNEKVLPKHSLLYEYFTVYNELTKVKYVTEGMRKPAFLSGEQKKAIVDLLFKTNRKVTVKQLKEDYFKKIECFDSVEISGVEDRFNASLGTYHDLLKIIKDKDFLDNEENEDILEDIVLTLTLFEDREMIEERLKTYAHLFDDKVMKQLKRRRYTGWGRLSRKLINGIRDKQSGKTILDFLKSDGFANRNFMQLIHDDSLTFKEDIQKAQVSGQGDSLHEHIANLAGSPAIKKGILQTVKVVDELVKVMGRHKPENIVIEMARENQTTQKGQKNSRERMKRIEEGIKELGSQILKEHPVENTQLQNEKLYLYYLQNGRDMYVDQELDINRLSDYDVDAIVPQSFLKDDSIDNKVLTRSDKNRGKSDNVPSEEVVKKMKNYWRQLLNAKLITQRKFDNLTKAERGGLSELDKAGFIKRQLVETRQITKHVAQILDSRMNTKYDENDKLIREVKVITLKSKLVSDFRKDFQFYKVREINNYHHAHDAYLNAVVGTALIKKYPKLESEFVYGDYKVYDVRKMIAKSEQEIGKATAKYFFYSNIMNFFKTEITLANGEIRKRPLIETNGETGEIVWDKGRDFATVRKVLSMPQVNIVKKTEVQTGGFSKESILPKRNSDKLIARKKDWDPKKYGGFDSPTVAYSVLVVAKVEKGKSKKLKSVKELLGITIMERSSFEKNPIDFLEAKGYKEVKKDLIIKLPKYSLFELENGRKRMLASAGELQKGNELALPSKYVNFLYLASHYEKLKGSPEDNEQKQLFVEQHKHYLDEIIEQISEFSKRVILADANLDKVLSAYNKHRDKPIREQAENIIHLFTLTNLGAPAAFKYFDTTIDRKRYTSTKEVLDATLIHQSITGLYETRIDLSQLGGDSRADypydvpdyaSGSpkkkrkvSPGggpssgapppsggspagsptsteegtsesatpesgpgtstepsegsapgspagsptsteegtstepsegsapgtstepseNHDQEFDPPKVYPPVPAEKRKPIRVLSLFDGIATGLLVLKDLGIQVDRYIASEVCEDSITVGMVRHQGKIMYVGDVRSVTQKHIQEWGPFDLVIGGSPCNDLSIVNPARKGLYEGTGRLFFEFYRLLHDARPKEGDDRPFFWLFENVVAMGVSDKRDISRFLESNPVMIDAKEVSAAHRARYFWGNLPGMNRPLASTVNDKLELQECLEHGRIAKFSKVRTITTRSNSIKQGKDQHFPVFMNEKEDILWCTEMERVFGFPVHYTDVSNMSRLARQRLLGRSWSVPVIRHLFAPLKEYFACVSSGNSNANSRGPSFSSGLVPLSLRGSHMGPMEIYKTVSAWKRQPVRVLSLFRNIDKVLKSLGFLESGSGSGGGTLKYVEDVTNVVRRDVEKWGPFDLVYGSTQPLGSSCDRCPGWYMFQFHRILQYALPRQESQRPFFWIFMDNLLLTEDDQETTTRFLQTEAVTLQDVRGRDYQNAMRVWSNIPGLKSKHAPLTPKEEEYLQAQVRSRSKLDAPKVDLLVKNCLLPLREYFKYFSQNSLPLSRADpkkkrkvGSGatnfsllkqagdveenpgpselikenmhmklymegtvdnhhfkctsegegkpyegtqtmrikvveggplpfafdilatsflygsktfinhtqgipdffkqsfpegftwervttyedggvltatqdtslqdgcliynvkirgvnftsngpvmqkktlgweaftetlypadgglegrndmalklvggshlianikttyrskkpaknlkmpgvyyvdyrlerikeannetyveqhevavarycdlpsklghkln *
[0283] SEQ ID NO: 12 (p100 (KRAB - XTEN16 - dCas9 - XTEN80 - Dnmt3A - Dnmt3L - P2A - P2A - BFP): KRAB (жирный шрифт, из Gilbert et al., Cell, 2013, 2014), XTEN16 (последовательность из 16 аминокислот), dCas9 (курсив), метка HA (строчные буквы), линкеры (подчеркивание), SV40 СЯЛ (строчные буквы, курсив), XTEN80 (строчные буквы, жирный шрифт, курсив, последовательность из 80 аминокислот), Dnmt3A (жирный шрифт, курсив, из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), линкер из 27 аминокислот (подчеркивание, курсив, из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), Dnmt3L (подчеркивание, жирный шрифт, из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), последовательность расщепления пептидом P2A (строчные буквы, жирный шрифт), BFP (строчные буквы, подчеркивание))
DAKSLTAWSRTLVTFKDVFVDFTREEWKLLDTAQQIVYRNVMLENYKNLVSLGYQLTKPDVILRLEKGEEPSGSETPGTSESATPESMDKKYSIGLAIGTNSVGWAVITDEYKVPSKKFKVLGNTDRHSIKKNLIGALLFDSGETAEATRLKRTARRRYTRRKNRICYLQEIFSNEMAKVDDSFFHRLEESFLVEEDKKHERHPIFGNIVDEVAYHEKYPTIYHLRKKLVDSTDKADLRLIYLALAHMIKFRGHFLIEGDLNPDNSDVDKLFIQLVQTYNQLFEENPINASGVDAKAILSARLSKSRRLENLIAQLPGEKKNGLFGNLIALSLGLTPNFKSNFDLAEDAKLQLSKDTYDDDLDNLLAQIGDQYADLFLAAKNLSDAILLSDILRVNTEITKAPLSASMIKRYDEHHQDLTLLKALVRQQLPEKYKEIFFDQSKNGYAGYIDGGASQEEFYKFIKPILEKMDGTEELLVKLNREDLLRKQRTFDNGSIPHQIHLGELHAILRRQEDFYPFLKDNREKIEKILTFRIPYYVGPLARGNSRFAWMTRKSEETITPWNFEEVVDKGASAQSFIERMTNFDKNLPNEKVLPKHSLLYEYFTVYNELTKVKYVTEGMRKPAFLSGEQKKAIVDLLFKTNRKVTVKQLKEDYFKKIECFDSVEISGVEDRFNASLGTYHDLLKIIKDKDFLDNEENEDILEDIVLTLTLFEDREMIEERLKTYAHLFDDKVMKQLKRRRYTGWGRLSRKLINGIRDKQSGKTILDFLKSDGFANRNFMQLIHDDSLTFKEDIQKAQVSGQGDSLHEHIANLAGSPAIKKGILQTVKVVDELVKVMGRHKPENIVIEMARENQTTQKGQKNSRERMKRIEEGIKELGSQILKEHPVENTQLQNEKLYLYYLQNGRDMYVDQELDINRLSDYDVDAIVPQSFLKDDSIDNKVLTRSDKNRGKSDNVPSEEVVKKMKNYWRQLLNAKLITQRKFDNLTKAERGGLSELDKAGFIKRQLVETRQITKHVAQILDSRMNTKYDENDKLIREVKVITLKSKLVSDFRKDFQFYKVREINNYHHAHDAYLNAVVGTALIKKYPKLESEFVYGDYKVYDVRKMIAKSEQEIGKATAKYFFYSNIMNFFKTEITLANGEIRKRPLIETNGETGEIVWDKGRDFATVRKVLSMPQVNIVKKTEVQTGGFSKESILPKRNSDKLIARKKDWDPKKYGGFDSPTVAYSVLVVAKVEKGKSKKLKSVKELLGITIMERSSFEKNPIDFLEAKGYKEVKKDLIIKLPKYSLFELENGRKRMLASAGELQKGNELALPSKYVNFLYLASHYEKLKGSPEDNEQKQLFVEQHKHYLDEIIEQISEFSKRVILADANLDKVLSAYNKHRDKPIREQAENIIHLFTLTNLGAPAAFKYFDTTIDRKRYTSTKEVLDATLIHQSITGLYETRIDLSQLGGDSRADypydvpdyaSGSpkkkrkvSPGggpssgapppsggspagsptsteegtsesatpesgpgtstepsegsapgspagsptsteegtstepsegsapgtstepseNHDQEFDPPKVYPPVPAEKRKPIRVLSLFDGIATGLLVLKDLGIQVDRYIASEVCEDSITVGMVRHQGKIMYVGDVRSVTQKHIQEWGPFDLVIGGSPCNDLSIVNPARKGLYEGTGRLFFEFYRLLHDARPKEGDDRPFFWLFENVVAMGVSDKRDISRFLESNPVMIDAKEVSAAHRARYFWGNLPGMNRPLASTVNDKLELQECLEHGRIAKFSKVRTITTRSNSIKQGKDQHFPVFMNEKEDILWCTEMERVFGFPVHYTDVSNMSRLARQRLLGRSWSVPVIRHLFAPLKEYFACVSSGNSNANSRGPSFSSGLVPLSLRGSHMGPMEIYKTVSAWKRQPVRVLSLFRNIDKVLKSLGFLESGSGSGGGTLKYVEDVTNVVRRDVEKWGPFDLVYGSTQPLGSSCDRCPGWYMFQFHRILQYALPRQESQRPFFWIFMDNLLLTEDDQETTTRFLQTEAVTLQDVRGRDYQNAMRVWSNIPGLKSKHAPLTPKEEEYLQAQVRSRSKLDAPKVDLLVKNCLLPLREYFKYFSQNSLPLSRADpkkkrkvGSGatnfsllkqagdveenpgpGSGatnfsllkqagdveenpgpselikenmhmklymegtvdnhhfkctsegegkpyegtqtmrikvveggplpfafdilatsflygsktfinhtqgipdffkqsfpegftwervttyedggvltatqdtslqdgcliynvkirgvnftsngpvmqkktlgweaftetlypadgglegrndmalklvggshlianikttyrskkpaknlkmpgvyyvdyrlerikeannetyveqhevavarycdlpsklghkln *
[0284] SEQ ID NO: 13 (p101 (KRAB - XTEN80 - dCas9 - XTEN16 - Dnmt3A - Dnmt3L - P2A - BFP): KRAB (жирный шрифт, из Gilbert et al., Cell, 2013, 2014), линкеры (подчеркивание), XTEN80 (строчные буквы, жирный шрифт, курсив, последовательность из 80 аминокислот), dCas9 (курсив), метка HA (строчные буквы), SV40 СЯЛ (строчные буквы, курсив), XTEN16 (последовательность из 16 аминокислот), Dnmt3A (жирный шрифт, курсив, из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), линкер из 27 аминокислот (подчеркивание, курсив, из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), Dnmt3L (подчеркивание, жирный шрифт, из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), последовательность расщепления пептидом P2A (строчные буквы, жирный шрифт), BFP (строчные буквы, подчеркивание))
DAKSLTAWSRTLVTFKDVFVDFTREEWKLLDTAQQIVYRNVMLENYKNLVSLGYQLTKPDVILRLEKGEEPggpssgapppsggspagsptsteegtsesatpesgpgtstepsegsapgspagsptsteegtstepsegsapgtstepse MDKKYSIGLAIGTNSVGWAVITDEYKVPSKKFKVLGNTDRHSIKKNLIGALLFDSGETAEATRLKRTARRRYTRRKNRICYLQEIFSNEMAKVDDSFFHRLEESFLVEEDKKHERHPIFGNIVDEVAYHEKYPTIYHLRKKLVDSTDKADLRLIYLALAHMIKFRGHFLIEGDLNPDNSDVDKLFIQLVQTYNQLFEENPINASGVDAKAILSARLSKSRRLENLIAQLPGEKKNGLFGNLIALSLGLTPNFKSNFDLAEDAKLQLSKDTYDDDLDNLLAQIGDQYADLFLAAKNLSDAILLSDILRVNTEITKAPLSASMIKRYDEHHQDLTLLKALVRQQLPEKYKEIFFDQSKNGYAGYIDGGASQEEFYKFIKPILEKMDGTEELLVKLNREDLLRKQRTFDNGSIPHQIHLGELHAILRRQEDFYPFLKDNREKIEKILTFRIPYYVGPLARGNSRFAWMTRKSEETITPWNFEEVVDKGASAQSFIERMTNFDKNLPNEKVLPKHSLLYEYFTVYNELTKVKYVTEGMRKPAFLSGEQKKAIVDLLFKTNRKVTVKQLKEDYFKKIECFDSVEISGVEDRFNASLGTYHDLLKIIKDKDFLDNEENEDILEDIVLTLTLFEDREMIEERLKTYAHLFDDKVMKQLKRRRYTGWGRLSRKLINGIRDKQSGKTILDFLKSDGFANRNFMQLIHDDSLTFKEDIQKAQVSGQGDSLHEHIANLAGSPAIKKGILQTVKVVDELVKVMGRHKPENIVIEMARENQTTQKGQKNSRERMKRIEEGIKELGSQILKEHPVENTQLQNEKLYLYYLQNGRDMYVDQELDINRLSDYDVDAIVPQSFLKDDSIDNKVLTRSDKNRGKSDNVPSEEVVKKMKNYWRQLLNAKLITQRKFDNLTKAERGGLSELDKAGFIKRQLVETRQITKHVAQILDSRMNTKYDENDKLIREVKVITLKSKLVSDFRKDFQFYKVREINNYHHAHDAYLNAVVGTALIKKYPKLESEFVYGDYKVYDVRKMIAKSEQEIGKATAKYFFYSNIMNFFKTEITLANGEIRKRPLIETNGETGEIVWDKGRDFATVRKVLSMPQVNIVKKTEVQTGGFSKESILPKRNSDKLIARKKDWDPKKYGGFDSPTVAYSVLVVAKVEKGKSKKLKSVKELLGITIMERSSFEKNPIDFLEAKGYKEVKKDLIIKLPKYSLFELENGRKRMLASAGELQKGNELALPSKYVNFLYLASHYEKLKGSPEDNEQKQLFVEQHKHYLDEIIEQISEFSKRVILADANLDKVLSAYNKHRDKPIREQAENIIHLFTLTNLGAPAAFKYFDTTIDRKRYTSTKEVLDATLIHQSITGLYETRIDLSQLGGD SRADypydvpdyaSGSpkkkrkvSPGSGSETPGTSESATPESNHDQEFDPPKVYPPVPAEKRKPIRVLSLFDGIATGLLVLKDLGIQVDRYIASEVCEDSITVGMVRHQGKIMYVGDVRSVTQKHIQEWGPFDLVIGGSPCNDLSIVNPARKGLYEGTGRLFFEFYRLLHDARPKEGDDRPFFWLFENVVAMGVSDKRDISRFLESNPVMIDAKEVSAAHRARYFWGNLPGMNRPLASTVNDKLELQECLEHGRIAKFSKVRTITTRSNSIKQGKDQHFPVFMNEKEDILWCTEMERVFGFPVHYTDVSNMSRLARQRLLGRSWSVPVIRHLFAPLKEYFACVSSGNSNANSRGPSFSSGLVPLSLRGSHMGPMEIYKTVSAWKRQPVRVLSLFRNIDKVLKSLGFLESGSGSGGGTLKYVEDVTNVVRRDVEKWGPFDLVYGSTQPLGSSCDRCPGWYMFQFHRILQYALPRQESQRPFFWIFMDNLLLTEDDQETTTRFLQTEAVTLQDVRGRDYQNAMRVWSNIPGLKSKHAPLTPKEEEYLQAQVRSRSKLDAPKVDLLVKNCLLPLREYFKYFSQNSLPLSRADpkkkrkvGSGatnfsllkqagdveenpgpselikenmhmklymegtvdnhhfkctsegegkpyegtqtmrikvveggplpfafdilatsflygsktfinhtqgipdffkqsfpegftwervttyedggvltatqdtslqdgcliynvkirgvnftsngpvmqkktlgweaftetlypadgglegrndmalklvggshlianikttyrskkpaknlkmpgvyyvdyrlerikeannetyveqhevavarycdlpsklghkln *
[0285] SEQ ID NO: 14 (p102 (KRAB - XTEN80 - dCas9 - XTEN16 - Dnmt3A - Dnmt3L - P2A - BFP): KRAB (жирный шрифт, из Gilbert et al., Cell, 2013, 2014), линкеры (подчеркивание), XTEN80 (строчные буквы, жирный шрифт, курсив, последовательность из 80 аминокислот), dCas9 (курсив), метка HA (строчные буквы), SV40 СЯЛ (строчные буквы, курсив), XTEN16 (последовательность из 16 аминокислот), Dnmt3A (жирный шрифт, курсив, из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), линкер из 27 аминокислот (подчеркивание, курсив, из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), Dnmt3L (подчеркивание, жирный шрифт, из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), последовательность расщепления пептидом P2A (строчные буквы, жирный шрифт), BFP (строчные буквы, подчеркивание))
DAKSLTAWSRTLVTFKDVFVDFTREEWKLLDTAQQIVYRNVMLENYKNLVSLGYQLTKPDVILRLEKGEEPggpssgapppsggspagsptsteegtsesatpesgpgtstepsegsapgspagsptsteegtstepsegsapgtstepse MDKKYSIGLAIGTNSVGWAVITDEYKVPSKKFKVLGNTDRHSIKKNLIGALLFDSGETAEATRLKRTARRRYTRRKNRICYLQEIFSNEMAKVDDSFFHRLEESFLVEEDKKHERHPIFGNIVDEVAYHEKYPTIYHLRKKLVDSTDKADLRLIYLALAHMIKFRGHFLIEGDLNPDNSDVDKLFIQLVQTYNQLFEENPINASGVDAKAILSARLSKSRRLENLIAQLPGEKKNGLFGNLIALSLGLTPNFKSNFDLAEDAKLQLSKDTYDDDLDNLLAQIGDQYADLFLAAKNLSDAILLSDILRVNTEITKAPLSASMIKRYDEHHQDLTLLKALVRQQLPEKYKEIFFDQSKNGYAGYIDGGASQEEFYKFIKPILEKMDGTEELLVKLNREDLLRKQRTFDNGSIPHQIHLGELHAILRRQEDFYPFLKDNREKIEKILTFRIPYYVGPLARGNSRFAWMTRKSEETITPWNFEEVVDKGASAQSFIERMTNFDKNLPNEKVLPKHSLLYEYFTVYNELTKVKYVTEGMRKPAFLSGEQKKAIVDLLFKTNRKVTVKQLKEDYFKKIECFDSVEISGVEDRFNASLGTYHDLLKIIKDKDFLDNEENEDILEDIVLTLTLFEDREMIEERLKTYAHLFDDKVMKQLKRRRYTGWGRLSRKLINGIRDKQSGKTILDFLKSDGFANRNFMQLIHDDSLTFKEDIQKAQVSGQGDSLHEHIANLAGSPAIKKGILQTVKVVDELVKVMGRHKPENIVIEMARENQTTQKGQKNSRERMKRIEEGIKELGSQILKEHPVENTQLQNEKLYLYYLQNGRDMYVDQELDINRLSDYDVDAIVPQSFLKDDSIDNKVLTRSDKNRGKSDNVPSEEVVKKMKNYWRQLLNAKLITQRKFDNLTKAERGGLSELDKAGFIKRQLVETRQITKHVAQILDSRMNTKYDENDKLIREVKVITLKSKLVSDFRKDFQFYKVREINNYHHAHDAYLNAVVGTALIKKYPKLESEFVYGDYKVYDVRKMIAKSEQEIGKATAKYFFYSNIMNFFKTEITLANGEIRKRPLIETNGETGEIVWDKGRDFATVRKVLSMPQVNIVKKTEVQTGGFSKESILPKRNSDKLIARKKDWDPKKYGGFDSPTVAYSVLVVAKVEKGKSKKLKSVKELLGITIMERSSFEKNPIDFLEAKGYKEVKKDLIIKLPKYSLFELENGRKRMLASAGELQKGNELALPSKYVNFLYLASHYEKLKGSPEDNEQKQLFVEQHKHYLDEIIEQISEFSKRVILADANLDKVLSAYNKHRDKPIREQAENIIHLFTLTNLGAPAAFKYFDTTIDRKRYTSTKEVLDATLIHQSITGLYETRIDLSQLGGD SRADypydvpdyaSGSpkkkrkvSPGSGSETPGTSESATPESNHDQEFDPPKVYPPVPAEKRKPIRVLSLFDGIATGLLVLKDLGIQVDRYIASEVCEDSITVGMVRHQGKIMYVGDVRSVTQKHIQEWGPFDLVIGGSPCNDLSIVNPARKGLYEGTGRLFFEFYRLLHDARPKEGDDRPFFWLFENVVAMGVSDKRDISRFLESNPVMIDAKEVSAAHRARYFWGNLPGMNRPLASTVNDKLELQECLEHGRIAKFSKVRTITTRSNSIKQGKDQHFPVFMNEKEDILWCTEMERVFGFPVHYTDVSNMSRLARQRLLGRSWSVPVIRHLFAPLKEYFACVSSGNSNANSRGPSFSSGLVPLSLRGSHMGPMEIYKTVSAWKRQPVRVLSLFRNIDKVLKSLGFLESGSGSGGGTLKYVEDVTNVVRRDVEKWGPFDLVYGSTQPLGSSCDRCPGWYMFQFHRILQYALPRQESQRPFFWIFMDNLLLTEDDQETTTRFLQTEAVTLQDVRGRDYQNAMRVWSNIPGLKSKHAPLTPKEEEYLQAQVRSRSKLDAPKVDLLVKNCLLPLREYFKYFSQNSLPLSRADpkkkrkvGSGatnfsllkqagdveenpgpGSGatnfsllkqagdveenpgpselikenmhmklymegtvdnhhfkctsegegkpyegtqtmrikvveggplpfafdilatsflygsktfinhtqgipdffkqsfpegftwervttyedggvltatqdtslqdgcliynvkirgvnftsngpvmqkktlgweaftetlypadgglegrndmalklvggshlianikttyrskkpaknlkmpgvyyvdyrlerikeannetyveqhevavarycdlpsklghkln *
[0286] SEQ ID NO: 15 (p112 (Dnmt3A - Dnmt3L - XTEN80 - dCas9 - BFP - KRAB); KRAB (жирный шрифт, из Gilbert et al., Cell, 2013, 2014), линкеры (подчеркивание), XTEN80 (строчные буквы, жирный шрифт, курсив, последовательность из 80 аминокислот), dCas9 (курсив), метка HA (строчные буквы), SV40 СЯЛ (строчные буквы, курсив), Dnmt3A (жирный шрифт, курсив, из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), линкер из 27 аминокислот (подчеркивание, курсив, из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), Dnmt3L (подчеркивание, жирный шрифт, из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016), BFP (строчные буквы, подчеркивание))
NHDQEFDPPKVYPPVPAEKRKPIRVLSLFDGIATGLLVLKDLGIQVDRYIASEVCEDSITVGMVRHQGKIMYVGDVRSVTQKHIQEWGPFDLVIGGSPCNDLSIVNPARKGLYEGTGRLFFEFYRLLHDARPKEGDDRPFFWLFENVVAMGVSDKRDISRFLESNPVMIDAKEVSAAHRARYFWGNLPGMNRPLASTVNDKLELQECLEHGRIAKFSKVRTITTRSNSIKQGKDQHFPVFMNEKEDILWCTEMERVFGFPVHYTDVSNMSRLARQRLLGRSWSVPVIRHLFAPLKEYFACV SSGNSNANSRGPSFSSGLVPLSLRGSH MGPMEIYKTVSAWKRQPVRVLSLFRNIDKVLKSLGFLESGSGSGGGTLKYVEDVTNVVRRDVEKWGPFDLVYGSTQPLGSSCDRCPGWYMFQFHRILQYALPRQESQRPFFWIFMDNLLLTEDDQETTTRFLQTEAVTLQDVRGRDYQNAMRVWSNIPGLKSKHAPLTPKEEEYLQAQVRSRSKLDAPKVDLLVKNCLLPLREYFKYFSQNSLPL ggpssgapppsggspagsptsteegtsesatpesgpgtstepsegsapgspagsptsteegtstepsegsapgtstepse MDKKYSIGLAIGTNSVGWAVITDEYKVPSKKFKVLGNTDRHSIKKNLIGALLFDSGETAEATRLKRTARRRYTRRKNRICYLQEIFSNEMAKVDDSFFHRLEESFLVEEDKKHERHPIFGNIVDEVAYHEKYPTIYHLRKKLVDSTDKADLRLIYLALAHMIKFRGHFLIEGDLNPDNSDVDKLFIQLVQTYNQLFEENPINASGVDAKAILSARLSKSRRLENLIAQLPGEKKNGLFGNLIALSLGLTPNFKSNFDLAEDAKLQLSKDTYDDDLDNLLAQIGDQYADLFLAAKNLSDAILLSDILRVNTEITKAPLSASMIKRYDEHHQDLTLLKALVRQQLPEKYKEIFFDQSKNGYAGYIDGGASQEEFYKFIKPILEKMDGTEELLVKLNREDLLRKQRTFDNGSIPHQIHLGELHAILRRQEDFYPFLKDNREKIEKILTFRIPYYVGPLARGNSRFAWMTRKSEETITPWNFEEVVDKGASAQSFIERMTNFDKNLPNEKVLPKHSLLYEYFTVYNELTKVKYVTEGMRKPAFLSGEQKKAIVDLLFKTNRKVTVKQLKEDYFKKIECFDSVEISGVEDRFNASLGTYHDLLKIIKDKDFLDNEENEDILEDIVLTLTLFEDREMIEERLKTYAHLFDDKVMKQLKRRRYTGWGRLSRKLINGIRDKQSGKTILDFLKSDGFANRNFMQLIHDDSLTFKEDIQKAQVSGQGDSLHEHIANLAGSPAIKKGILQTVKVVDELVKVMGRHKPENIVIEMARENQTTQKGQKNSRERMKRIEEGIKELGSQILKEHPVENTQLQNEKLYLYYLQNGRDMYVDQELDINRLSDYDVDAIVPQSFLKDDSIDNKVLTRSDKNRGKSDNVPSEEVVKKMKNYWRQLLNAKLITQRKFDNLTKAERGGLSELDKAGFIKRQLVETRQITKHVAQILDSRMNTKYDENDKLIREVKVITLKSKLVSDFRKDFQFYKVREINNYHHAHDAYLNAVVGTALIKKYPKLESEFVYGDYKVYDVRKMIAKSEQEIGKATAKYFFYSNIMNFFKTEITLANGEIRKRPLIETNGETGEIVWDKGRDFATVRKVLSMPQVNIVKKTEVQTGGFSKESILPKRNSDKLIARKKDWDPKKYGGFDSPTVAYSVLVVAKVEKGKSKKLKSVKELLGITIMERSSFEKNPIDFLEAKGYKEVKKDLIIKLPKYSLFELENGRKRMLASAGELQKGNELALPSKYVNFLYLASHYEKLKGSPEDNEQKQLFVEQHKHYLDEIIEQISEFSKRVILADANLDKVLSAYNKHRDKPIREQAENIIHLFTLTNLGAPAAFKYFDTTIDRKRYTSTKEVLDATLIHQSITGLYETRIDLSQLGGD AypydvpdyaSLGSGSpkkkrkvEDpkkkrkvDGIGSGSNGSSGSselikenmhmklymegtvdnhhfkctsegegkpyegtqtmrikvveggplpfafdilatsflygsktfinhtqgipdffkqsfpegftwervttyedggvltatqdtslqdgcliynvkirgvnftsngpvmqkktlgweaftetlypadgglegrndmalklvggshlianikttyrskkpaknlkmpgvyyvdyrlerikeannetyveqhevavarycdlpsklghklnGGGGGMDAKSLTAWSRTLVTFKDVFVDFTREEWKLLDTAQQIVYRNVMLENYKNLVSLGYQLTKPDVILRLEKGEEP *
[0287] SEQ ID NO: 16 (KRAB; из Gilbert et al., Cell, 2013, 2014)
DAKSLTAWSRTLVTFKDVFVDFTREEWKLLDTAQQIVYRNVMLENYKNLVSLGYQLTKPDVILRLEKGEEP
[0288] SEQ ID NO: 17 (линкер)
GGSGGGS
[0289] SEQ ID NO: 18 (линкер)
SGS
[0290] SEQ ID NO: 19 (линкер)
EASGSGRASPGIPGSTR
[0291] SEQ ID NO: 20 (линкер)
SRAD
[0292] SEQ ID NO: 21 (линкер)
GSG
[0293] SEQ ID NO: 22 (линкер
SPG
[0294] SEQ ID NO: 23 (dCas9)
MDKKYSIGLAIGTNSVGWAVITDEYKVPSKKFKVLGNTDRHSIKKNLIGALLFDSGETAEATRLKRTARRRYTRRKNRICYLQEIFSNEMAKVDDSFFHRLEESFLVEEDKKHERHPIFGNIVDEVAYHEKYPTIYHLRKKLVDSTDKADLRLIYLALAHMIKFRGHFLIEGDLNPDNSDVDKLFIQLVQTYNQLFEENPINASGVDAKAILSARLSKSRRLENLIAQLPGEKKNGLFGNLIALSLGLTPNFKSNFDLAEDAKLQLSKDTYDDDLDNLLAQIGDQYADLFLAAKNLSDAILLSDILRVNTEITKAPLSASMIKRYDEHHQDLTLLKALVRQQLPEKYKEIFFDQSKNGYAGYIDGGASQEEFYKFIKPILEKMDGTEELLVKLNREDLLRKQRTFDNGSIPHQIHLGELHAILRRQEDFYPFLKDNREKIEKILTFRIPYYVGPLARGNSRFAWMTRKSEETITPWNFEEVVDKGASAQSFIERMTNFDKNLPNEKVLPKHSLLYEYFTVYNELTKVKYVTEGMRKPAFLSGEQKKAIVDLLFKTNRKVTVKQLKEDYFKKIECFDSVEISGVEDRFNASLGTYHDLLKIIKDKDFLDNEENEDILEDIVLTLTLFEDREMIEERLKTYAHLFDDKVMKQLKRRRYTGWGRLSRKLINGIRDKQSGKTILDFLKSDGFANRNFMQLIHDDSLTFKEDIQKAQVSGQGDSLHEHIANLAGSPAIKKGILQTVKVVDELVKVMGRHKPENIVIEMARENQTTQKGQKNSRERMKRIEEGIKELGSQILKEHPVENTQLQNEKLYLYYLQNGRDMYVDQELDINRLSDYDVDAIVPQSFLKDDSIDNKVLTRSDKNRGKSDNVPSEEVVKKMKNYWRQLLNAKLITQRKFDNLTKAERGGLSELDKAGFIKRQLVETRQITKHVAQILDSRMNTKYDENDKLIREVKVITLKSKLVSDFRKDFQFYKVREINNYHHAHDAYLNAVVGTALIKKYPKLESEFVYGDYKVYDVRKMIAKSEQEIGKATAKYFFYSNIMNFFKTEITLANGEIRKRPLIETNGETGEIVWDKGRDFATVRKVLSMPQVNIVKKTEVQTGGFSKESILPKRNSDKLIARKKDWDPKKYGGFDSPTVAYSVLVVAKVEKGKSKKLKSVKELLGITIMERSSFEKNPIDFLEAKGYKEVKKDLIIKLPKYSLFELENGRKRMLASAGELQKGNELALPSKYVNFLYLASHYEKLKGSPEDNEQKQLFVEQHKHYLDEIIEQISEFSKRVILADANLDKVLSAYNKHRDKPIREQAENIIHLFTLTNLGAPAAFKYFDTTIDRKRYTSTKEVLDATLIHQSITGLYETRIDLSQLGGD
[0295] SEQ ID NO: 24 (метка НА)
YPYDVPDYA
[0296] SEQ ID NO: 25 (SV40 СЯЛ)
PKKKRKV
[0297] SEQ ID NO: 26 (Dnmt3A; остатки 612-912; из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016)
NHDQEFDPPKVYPPVPAEKRKPIRVLSLFDGIATGLLVLKDLGIQVDRYIASEVCEDSITVGMVRHQGKIMYVGDVRSVTQKHIQEWGPFDLVIGGSPCNDLSIVNPARKGLYEGTGRLFFEFYRLLHDARPKEGDDRPFFWLFENVVAMGVSDKRDISRFLESNPVMIDAKEVSAAHRARYFWGNLPGMNRPLASTVNDKLELQECLEHGRIAKFSKVRTITTRSNSIKQGKDQHFPVFMNEKEDILWCTEMERVFGFPVHYTDVSNMSRLARQRLLGRSWSVPVIRHLFAPLKEYFACV
[0298] SEQ ID NO: 27 (линкер из 27 аминокислот; из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016)
SSGNSNANSRGPSFSSGLVPLSLRGSH
[0299] SEQ ID NO: 28 (Dnmt3L; из Siddique et al., JMB, 2013; Stepper et al., NAR, 2016)
MGPMEIYKTVSAWKRQPVRVLSLFRNIDKVLKSLGFLESGSGSGGGTLKYVEDVTNVVRRDVEKWGPFDLVYGSTQPLGSSCDRCPGWYMFQFHRILQYALPRQESQRPFFWIFMDNLLLTEDDQETTTRFLQTEAVTLQDVRGRDYQNAMRVWSNIPGLKSKHAPLTPKEEEYLQAQVRSRSKLDAPKVDLLVKNCLLPLREYFKYFSQNSLPL
[0300] SEQ ID NO: 29 (последовательность расщепления пептидом P2A)
ATNFSLLKQAGDVEENPGP
[0301] SEQ ID NO: 30 (СФП, англ. «BFP»)
SELIKENMHMKLYMEGTVDNHHFKCTSEGEGKPYEGTQTMRIKVVEGGPLPFAFDILATSFLYGSKTFINHTQGIPDFFKQSFPEGFTWERVTTYEDGGVLTATQDTSLQDGCLIYNVKIRGVNFTSNGPVMQKKTLGWEAFTETLYPADGGLEGRNDMALKLVGGSHLIANIKTTYRSKKPAKNLKMPGVYYVDYRLERIKEANNETYVEQHEVAVARYCDLPSKLGHKLN *
[0302] SEQ ID NO: 31 (XTEN16 (последовательность из 16 аминокислот))
SGSETPGTSESATPES
[0303] SEQ ID NO: 32 (XTEN80 (последовательность из 80 аминокислот))
GGPSSGAPPPSGGSPAGSPTSTEEGTSESATPESGPGTSTEPSEGSAPGSPAGSPTSTEEGTSTEPSEGSAPGTSTEPSE
[0304] SEQ ID NO: 33 (домен Dnmt3A - Dnmt3L)
NHDQEFDPPKVYPPVPAEKRKPIRVLSLFDGIATGLLVLKDLGIQVDRYIASEVCEDSITVGMVRHQGKIMYVGDVRSVTQKHIQEWGPFDLVIGGSPCNDLSIVNPARKGLYEGTGRLFFEFYRLLHDARPKEGDDRPFFWLFENVVAMGVSDKRDISRFLESNPVMIDAKEVSAAHRARYFWGNLPGMNRPLASTVNDKLELQECLEHGRIAKFSKVRTITTRSNSIKQGKDQHFPVFMNEKEDILWCTEMERVFGFPVHYTDVSNMSRLARQRLLGRSWSVPVIRHLFAPLKEYFACVSSGNSNANSRGPSFSSGLVPLSLRGSHMGPMEIYKTVSAWKRQPVRVLSLFRNIDKVLKSLGFLESGSGSGGGTLKYVEDVTNVVRRDVEKWGPFDLVYGSTQPLGSSCDRCPGWYMFQFHRILQYALPRQESQRPFFWIFMDNLLLTEDDQETTTRFLQTEAVTLQDVRGRDYQNAMRVWSNIPGLKSKHAPLTPKEEEYLQAQVRSRSKLDAPKVDLLVKNCLLPLREYFKYFSQNSLPL
[0305] SEQ ID NO: 34 (ddAsCfp1)
MTQFEGFTNLYQVSKTLRFELIPQGKTLKHIQEQGFIEEDKARNDHYKELKPIIDRIYKTYADQCLQLVQLDWENLSAAIDSYRKEKTEETRNALIEEQATYRNAIHDYFIGRTDNLTDAINKRHAEIYKGLFKAELFNGKVLKQLGTVTTTEHENALLRSFDKFTTYFSGFYENRKNVFSAEDISTAIPHRIVQDNFPKFKENCHIFTRLITAVPSLREHFENVKKAIGIFVSTSIEEVFSFPFYNQLLTQTQIDLYNQLLGGISREAGTEKIKGLNEVLNLAIQKNDETAHIIASLPHRFIPLFKQILSDRNTLSFILEEFKSDEEVIQSFCKYKTLLRNENVLETAEALFNELNSIDLTHIFISHKKLETISSALCDHWDTLRNALYERRISELTGKITKSAKEKVQRSLKHEDINLQEIISAAGKELSEAFKQKTSEILSHAHAALDQPLPTTLKKQEEKEILKSQLDSLLGLYHLLDWFAVDESNEVDPEFSARLTGIKLEMEPSLSFYNKARNYATKKPYSVEKFKLNFQMPTLASGWDVNKEKNNGAILFVKNGLYYLGIMPKQKGRYKALSFEPTEKTSEGFDKMYYDYFPDAAKMIPKCSTQLKAVTAHFQTHTTPILLSNNFIEPLEITKEIYDLNNPEKEPKKFQTAYAKKTGDQKGYREALCKWIDFTRDFLSKYTKTTSIDLSSLRPSSQYKDLGEYYAELNPLLYHISFQRIAEKEIMDAVETGKLYLFQIYNKDFAKGHHGKPNLHTLYWTGLFSPENLAKTSIKLNGQAELFYRPKSRMKRMAHRLGEKMLNKKLKDQKTPIPDTLYQELYDYVNHRLSHDLSDEARALLPNVITKEVSHEIIKDRRFTSDKFFFHVPITLNYQAANSPSKFNQRVNAYLKEHPETPIIGIDRGERNLIYITVIDSTGKILEQRSLNTIQQFDYQKKLDNREKERVAARQAWSVVGTIKDLKQGYLSQVIHEIVDLMIHYQAVVVLANLNFGFKSKRTGIAEKAVYQQFEKMLIDKLNCLVLKDYPAEKVGGVLNPYQLTDQFTSFAKMGTQSGFLFYVPAPYTSKIDPLTGFVDPFVWKTIKNHESRKHFLEGFDFLHYDVKTGDFILHFKMNRNLSFQRGLPGFMPAWDIVFEKNETQFDAKGTPFIAGKRIVPVIENHRFTGRYRDLYPANELIALLEEKGIVFRDGSNILPKLLENDDSHAIDTMVALIRSVLQMRNSNAATGEDYINSPVRDLNGVCFDSRFQNPEWPMDADANGAYHIALKGQLLLNHLKESKDLKLQNGISNQDWLAYIQELRN
[0306] SEQ ID NO: 35 (ddLbCfp1)
MSKLEKFTNCYSLSKTLRFKAIPVGKTQENIDNKRLLVEDEKRAEDYKGVKKLLDRYYLSFINDVLHSIKLKNLNNYISLFRKKTRTEKENKELENLEINLRKEIAKAFKGNEGYKSLFKKDIIETILPEFLDDKDEIALVNSFNGFTTAFTGFFDNRENMFSEEAKSTSIAFRCINENLTRYISNMDIFEKVDAIFDKHEVQEIKEKILNSDYDVEDFFEGEFFNFVLTQEGIDVYNAIIGGFVTESGEKIKGLNEYINLYNQKTKQKLPKFKPLYKQVLSDRESLSFYGEGYTSDEEVLEVFRNTLNKNSEIFSSIKKLEKLFKNFDEYSSAGIFVKNGPAISTISKDIFGEWNVIRDKWNAEYDDIHLKKKAVVTEKYEDDRRKSFKKIGSFSLEQLQEYADADLSVVEKLKEIIIQKVDEIYKVYGSSEKLFDADFVLEKSLKKNDAVVAIMKDLLDSVKSFENYIKAFFGEGKETNRDESFYGDFVLAYDILLKVDHIYDAIRNYVTQKPYSKDKFKLYFQNPQFMGGWDKDKETDYRATILRYGSKYYLAIMDKKYAKCLQKIDKDDVNGNYEKINYKLLPGPNKMLPKVFFSKKWMAYYNPSEDIQKIYKNGTFKKGDMFNLNDCHKLIDFFKDSISRYPKWSNAYDFNFSETEKYKDIAGFYREVEEQGYKVSFESASKKEVDKLVEEGKLYMFQIYNKDFSDKSHGTPNLHTMYFKLLFDENNHGQIRLSGGAELFMRRASLKKEELVVHPANSPIANKNPDNPKKTTTLSYDVYKDKRFSEDQYELHIPIAINKCPKNIFKINTEVRVLLKHDDNPYVIGIARGERNLLYIVVVDGKGNIVEQYSLNEIINNFNGIRIKTDYHSLLDKKEKERFEARQNWTSIENIKELKAGYISQVVHKICELVEKYDAVIALADLNSGFKNSRVKVEKQVYQKFEKMLIDKLNYMVDKKSNPCATGGALKGYQITNKFESFKSMSTQNGFIFYIPAWLTSKIDPSTGFVNLLKTKYTSIADSKKFISSFDRIMYVPEEDLFEFALDYKNFSRTDADYIKKWKLYSYGNRIRIFRNPKKNNVFDWEEVCLTSAYKELFNKYGINYQQGDIRALLCEQSDKAFYSSFMALMSLMLQMRNSITGRTDVDFLISPVKNSDGIFYDSRNYEAQENAILPKNADANGAYNIARKVLWAIGQFKKAEDEKLDKVKIAISNKEWLEYAQTSVKH
[0307] SEQ ID NO: 36 (ddFnCfp1)
MYPYDVPDYASGSGMSIYQEFVNKYSLSKTLRFELIPQGKTLENIKARGLILDDEKRAKDYKKAKQIIDKYHQFFIEEILSSVCISEDLLQNYSDVYFKLKKSDDDNLQKDFKSAKDTIKKQISEYIKDSEKFKNLFNQNLIDAKKGQESDLILWLKQSKDNGIELFKANSDITDIDEALEIIKSFKGWTTYFKGFHENRKNVYSSNDIPTSIIYRIVDDNLPKFLENKAKYESLKDKAPEAINYEQIKKDLAEELTFDIDYKTSEVNQRVFSLDEVFEIANFNNYLNQSGITKFNTIIGGKFVNGENTKRKGINEYINLYSQQINDKTLKKYKMSVLFKQILSDTESKSFVIDKLEDDSDVVTTMQSFYEQIAAFKTVEEKSIKETLSLLFDDLKAQKLDLSKIYFKNDKSLTDLSQQVFDDYSVIGTAVLEYITQQIAPKNLDNPSKKEQELIAKKTEKAKYLSLETIKLALEEFNKHRDIDKQCRFEEILANFAAIPMIFDEIAQNKDNLAQISIKYQNQGKKDLLQASAEDDVKAIKDLLDQTNNLLHKLKIFHISQSEDKANILDKDEHFYLVFEECYFELANIVPLYNKIRNYITQKPYSDEKFKLNFENSTLANGWDKNKEPDNTAILFIKDDKYYLGVMNKKNNKIFDDKAIKENKGEGYKKIVYKLLPGANKMLPKVFFSAKSIKFYNPSEDILRIRNHSTHTKNGSPQKGYEKFEFNIEDCRKFIDFYKQSISKHPEWKDFGFRFSDTQRYNSIDEFYREVENQGYKLTFENISESYIDSVVNQGKLYLFQIYNKDFSAYSKGRPNLHTLYWKALFDERNLQDVVYKLNGEAELFYRKQSIPKKITHPAKEAIANKNKDNPKKESVFEYDLIKDKRFTEDKFFFHCPITINFKSSGANKFNDEINLLLKEKANDVHILSIARGERHLAYYTLVDGKGNIIKQDTFNIIGNDRMKTNYHDKLAAIEKDRDSARKDWKKINNIKEMKEGYLSQVVHEIAKLVIEYNAIVVFEDLNFGFKRGRFKVEKQVYQKLEKMLIEKLNYLVFKDNEFDKTGGVLRAYQLTAPFETFKKMGKQTGIIYYVPAGFTSKICPVTGFVNQLYPKYESVSKSQEFFSKFDKICYNLDKGYFEFSFDYKNFGDKAAKGKWTIASFGSRLINFRNSDKNHNWDTREVYPTKELEKLLKDYSIEYGHGECIKAAICGESDKKFFAKLTSVLNTILQMRNSKTGTELDYLISPVADVNGNFFDSRQAPKNMPQDADANGAYHIGLKGLMLLGRIKNNQEGKKLNLVIKNEEYFEFVQNRNN
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> The Regents of the University of California
Chen, Jin
Gilbert, Luke
Nunez, James
Weissman, Jonathan
<120> Композиции и способы редактирования генов
<130> 048536-620001WO
<140> PCT/US2019/028377
<141> 2019-04-19
<150> US 62/660,023
<151> 2018-04-19
<160> 96
<170> PatentIn, версия 3.5
<210> 1
<211> 2294
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетический полипептид
<400> 1
Asp Ala Lys Ser Leu Thr Ala Trp Ser Arg Thr Leu Val Thr Phe Lys
1. 5 10 15
Asp Val Phe Val Asp Phe Thr Arg Glu Glu Trp Lys Leu Leu Asp Thr
20 25 30
Ala Gln Gln Ile Val Tyr Arg Asn Val Met Leu Glu Asn Tyr Lys Asn
35 40 45
Leu Val Ser Leu Gly Tyr Gln Leu Thr Lys Pro Asp Val Ile Leu Arg
50 55 60
Leu Glu Lys Gly Glu Glu Pro Gly Gly Ser Gly Gly Gly Ser Met Asp
65 70 75 80
Lys Lys Tyr Ser Ile Gly Leu Ala Ile Gly Thr Asn Ser Val Gly Trp
85 90 95
Ala Val Ile Thr Asp Glu Tyr Lys Val Pro Ser Lys Lys Phe Lys Val
100 105 110
Leu Gly Asn Thr Asp Arg His Ser Ile Lys Lys Asn Leu Ile Gly Ala
115 120 125
Leu Leu Phe Asp Ser Gly Glu Thr Ala Glu Ala Thr Arg Leu Lys Arg
130 135 140
Thr Ala Arg Arg Arg Tyr Thr Arg Arg Lys Asn Arg Ile Cys Tyr Leu
145 150 155 160
Gln Glu Ile Phe Ser Asn Glu Met Ala Lys Val Asp Asp Ser Phe Phe
165 170 175
His Arg Leu Glu Glu Ser Phe Leu Val Glu Glu Asp Lys Lys His Glu
180 185 190
Arg His Pro Ile Phe Gly Asn Ile Val Asp Glu Val Ala Tyr His Glu
195 200 205
Lys Tyr Pro Thr Ile Tyr His Leu Arg Lys Lys Leu Val Asp Ser Thr
210 215 220
Asp Lys Ala Asp Leu Arg Leu Ile Tyr Leu Ala Leu Ala His Met Ile
225 230 235 240
Lys Phe Arg Gly His Phe Leu Ile Glu Gly Asp Leu Asn Pro Asp Asn
245 250 255
Ser Asp Val Asp Lys Leu Phe Ile Gln Leu Val Gln Thr Tyr Asn Gln
260 265 270
Leu Phe Glu Glu Asn Pro Ile Asn Ala Ser Gly Val Asp Ala Lys Ala
275 280 285
Ile Leu Ser Ala Arg Leu Ser Lys Ser Arg Arg Leu Glu Asn Leu Ile
290 295 300
Ala Gln Leu Pro Gly Glu Lys Lys Asn Gly Leu Phe Gly Asn Leu Ile
305 310 315 320
Ala Leu Ser Leu Gly Leu Thr Pro Asn Phe Lys Ser Asn Phe Asp Leu
325 330 335
Ala Glu Asp Ala Lys Leu Gln Leu Ser Lys Asp Thr Tyr Asp Asp Asp
340 345 350
Leu Asp Asn Leu Leu Ala Gln Ile Gly Asp Gln Tyr Ala Asp Leu Phe
355 360 365
Leu Ala Ala Lys Asn Leu Ser Asp Ala Ile Leu Leu Ser Asp Ile Leu
370 375 380
Arg Val Asn Thr Glu Ile Thr Lys Ala Pro Leu Ser Ala Ser Met Ile
385 390 395 400
Lys Arg Tyr Asp Glu His His Gln Asp Leu Thr Leu Leu Lys Ala Leu
405 410 415
Val Arg Gln Gln Leu Pro Glu Lys Tyr Lys Glu Ile Phe Phe Asp Gln
420 425 430
Ser Lys Asn Gly Tyr Ala Gly Tyr Ile Asp Gly Gly Ala Ser Gln Glu
435 440 445
Glu Phe Tyr Lys Phe Ile Lys Pro Ile Leu Glu Lys Met Asp Gly Thr
450 455 460
Glu Glu Leu Leu Val Lys Leu Asn Arg Glu Asp Leu Leu Arg Lys Gln
465 470 475 480
Arg Thr Phe Asp Asn Gly Ser Ile Pro His Gln Ile His Leu Gly Glu
485 490 495
Leu His Ala Ile Leu Arg Arg Gln Glu Asp Phe Tyr Pro Phe Leu Lys
500 505 510
Asp Asn Arg Glu Lys Ile Glu Lys Ile Leu Thr Phe Arg Ile Pro Tyr
515 520 525
Tyr Val Gly Pro Leu Ala Arg Gly Asn Ser Arg Phe Ala Trp Met Thr
530 535 540
Arg Lys Ser Glu Glu Thr Ile Thr Pro Trp Asn Phe Glu Glu Val Val
545 550 555 560
Asp Lys Gly Ala Ser Ala Gln Ser Phe Ile Glu Arg Met Thr Asn Phe
565 570 575
Asp Lys Asn Leu Pro Asn Glu Lys Val Leu Pro Lys His Ser Leu Leu
580 585 590
Tyr Glu Tyr Phe Thr Val Tyr Asn Glu Leu Thr Lys Val Lys Tyr Val
595 600 605
Thr Glu Gly Met Arg Lys Pro Ala Phe Leu Ser Gly Glu Gln Lys Lys
610 615 620
Ala Ile Val Asp Leu Leu Phe Lys Thr Asn Arg Lys Val Thr Val Lys
625 630 635 640
Gln Leu Lys Glu Asp Tyr Phe Lys Lys Ile Glu Cys Phe Asp Ser Val
645 650 655
Glu Ile Ser Gly Val Glu Asp Arg Phe Asn Ala Ser Leu Gly Thr Tyr
660 665 670
His Asp Leu Leu Lys Ile Ile Lys Asp Lys Asp Phe Leu Asp Asn Glu
675 680 685
Glu Asn Glu Asp Ile Leu Glu Asp Ile Val Leu Thr Leu Thr Leu Phe
690 695 700
Glu Asp Arg Glu Met Ile Glu Glu Arg Leu Lys Thr Tyr Ala His Leu
705 710 715 720
Phe Asp Asp Lys Val Met Lys Gln Leu Lys Arg Arg Arg Tyr Thr Gly
725 730 735
Trp Gly Arg Leu Ser Arg Lys Leu Ile Asn Gly Ile Arg Asp Lys Gln
740 745 750
Ser Gly Lys Thr Ile Leu Asp Phe Leu Lys Ser Asp Gly Phe Ala Asn
755 760 765
Arg Asn Phe Met Gln Leu Ile His Asp Asp Ser Leu Thr Phe Lys Glu
770 775 780
Asp Ile Gln Lys Ala Gln Val Ser Gly Gln Gly Asp Ser Leu His Glu
785 790 795 800
His Ile Ala Asn Leu Ala Gly Ser Pro Ala Ile Lys Lys Gly Ile Leu
805 810 815
Gln Thr Val Lys Val Val Asp Glu Leu Val Lys Val Met Gly Arg His
820 825 830
Lys Pro Glu Asn Ile Val Ile Glu Met Ala Arg Glu Asn Gln Thr Thr
835 840 845
Gln Lys Gly Gln Lys Asn Ser Arg Glu Arg Met Lys Arg Ile Glu Glu
850 855 860
Gly Ile Lys Glu Leu Gly Ser Gln Ile Leu Lys Glu His Pro Val Glu
865 870 875 880
Asn Thr Gln Leu Gln Asn Glu Lys Leu Tyr Leu Tyr Tyr Leu Gln Asn
885 890 895
Gly Arg Asp Met Tyr Val Asp Gln Glu Leu Asp Ile Asn Arg Leu Ser
900 905 910
Asp Tyr Asp Val Asp Ala Ile Val Pro Gln Ser Phe Leu Lys Asp Asp
915 920 925
Ser Ile Asp Asn Lys Val Leu Thr Arg Ser Asp Lys Asn Arg Gly Lys
930 935 940
Ser Asp Asn Val Pro Ser Glu Glu Val Val Lys Lys Met Lys Asn Tyr
945 950 955 960
Trp Arg Gln Leu Leu Asn Ala Lys Leu Ile Thr Gln Arg Lys Phe Asp
965 970 975
Asn Leu Thr Lys Ala Glu Arg Gly Gly Leu Ser Glu Leu Asp Lys Ala
980 985 990
Gly Phe Ile Lys Arg Gln Leu Val Glu Thr Arg Gln Ile Thr Lys His
995 1000 1005
Val Ala Gln Ile Leu Asp Ser Arg Met Asn Thr Lys Tyr Asp Glu
1010 1015 1020
Asn Asp Lys Leu Ile Arg Glu Val Lys Val Ile Thr Leu Lys Ser
1025 1030 1035
Lys Leu Val Ser Asp Phe Arg Lys Asp Phe Gln Phe Tyr Lys Val
1040 1045 1050
Arg Glu Ile Asn Asn Tyr His His Ala His Asp Ala Tyr Leu Asn
1055 1060 1065
Ala Val Val Gly Thr Ala Leu Ile Lys Lys Tyr Pro Lys Leu Glu
1070 1075 1080
Ser Glu Phe Val Tyr Gly Asp Tyr Lys Val Tyr Asp Val Arg Lys
1085 1090 1095
Met Ile Ala Lys Ser Glu Gln Glu Ile Gly Lys Ala Thr Ala Lys
1100 1105 1110
Tyr Phe Phe Tyr Ser Asn Ile Met Asn Phe Phe Lys Thr Glu Ile
1115 1120 1125
Thr Leu Ala Asn Gly Glu Ile Arg Lys Arg Pro Leu Ile Glu Thr
1130 1135 1140
Asn Gly Glu Thr Gly Glu Ile Val Trp Asp Lys Gly Arg Asp Phe
1145 1150 1155
Ala Thr Val Arg Lys Val Leu Ser Met Pro Gln Val Asn Ile Val
1160 1165 1170
Lys Lys Thr Glu Val Gln Thr Gly Gly Phe Ser Lys Glu Ser Ile
1175 1180 1185
Leu Pro Lys Arg Asn Ser Asp Lys Leu Ile Ala Arg Lys Lys Asp
1190 1195 1200
Trp Asp Pro Lys Lys Tyr Gly Gly Phe Asp Ser Pro Thr Val Ala
1205 1210 1215
Tyr Ser Val Leu Val Val Ala Lys Val Glu Lys Gly Lys Ser Lys
1220 1225 1230
Lys Leu Lys Ser Val Lys Glu Leu Leu Gly Ile Thr Ile Met Glu
1235 1240 1245
Arg Ser Ser Phe Glu Lys Asn Pro Ile Asp Phe Leu Glu Ala Lys
1250 1255 1260
Gly Tyr Lys Glu Val Lys Lys Asp Leu Ile Ile Lys Leu Pro Lys
1265 1270 1275
Tyr Ser Leu Phe Glu Leu Glu Asn Gly Arg Lys Arg Met Leu Ala
1280 1285 1290
Ser Ala Gly Glu Leu Gln Lys Gly Asn Glu Leu Ala Leu Pro Ser
1295 1300 1305
Lys Tyr Val Asn Phe Leu Tyr Leu Ala Ser His Tyr Glu Lys Leu
1310 1315 1320
Lys Gly Ser Pro Glu Asp Asn Glu Gln Lys Gln Leu Phe Val Glu
1325 1330 1335
Gln His Lys His Tyr Leu Asp Glu Ile Ile Glu Gln Ile Ser Glu
1340 1345 1350
Phe Ser Lys Arg Val Ile Leu Ala Asp Ala Asn Leu Asp Lys Val
1355 1360 1365
Leu Ser Ala Tyr Asn Lys His Arg Asp Lys Pro Ile Arg Glu Gln
1370 1375 1380
Ala Glu Asn Ile Ile His Leu Phe Thr Leu Thr Asn Leu Gly Ala
1385 1390 1395
Pro Ala Ala Phe Lys Tyr Phe Asp Thr Thr Ile Asp Arg Lys Arg
1400 1405 1410
Tyr Thr Ser Thr Lys Glu Val Leu Asp Ala Thr Leu Ile His Gln
1415 1420 1425
Ser Ile Thr Gly Leu Tyr Glu Thr Arg Ile Asp Leu Ser Gln Leu
1430 1435 1440
Gly Gly Asp Ser Arg Ala Asp Tyr Pro Tyr Asp Val Pro Asp Tyr
1445 1450 1455
Ala Ser Gly Ser Pro Lys Lys Lys Arg Lys Val Glu Ala Ser Gly
1460 1465 1470
Ser Gly Arg Ala Ser Pro Gly Ile Pro Gly Ser Thr Arg Asn His
1475 1480 1485
Asp Gln Glu Phe Asp Pro Pro Lys Val Tyr Pro Pro Val Pro Ala
1490 1495 1500
Glu Lys Arg Lys Pro Ile Arg Val Leu Ser Leu Phe Asp Gly Ile
1505 1510 1515
Ala Thr Gly Leu Leu Val Leu Lys Asp Leu Gly Ile Gln Val Asp
1520 1525 1530
Arg Tyr Ile Ala Ser Glu Val Cys Glu Asp Ser Ile Thr Val Gly
1535 1540 1545
Met Val Arg His Gln Gly Lys Ile Met Tyr Val Gly Asp Val Arg
1550 1555 1560
Ser Val Thr Gln Lys His Ile Gln Glu Trp Gly Pro Phe Asp Leu
1565 1570 1575
Val Ile Gly Gly Ser Pro Cys Asn Asp Leu Ser Ile Val Asn Pro
1580 1585 1590
Ala Arg Lys Gly Leu Tyr Glu Gly Thr Gly Arg Leu Phe Phe Glu
1595 1600 1605
Phe Tyr Arg Leu Leu His Asp Ala Arg Pro Lys Glu Gly Asp Asp
1610 1615 1620
Arg Pro Phe Phe Trp Leu Phe Glu Asn Val Val Ala Met Gly Val
1625 1630 1635
Ser Asp Lys Arg Asp Ile Ser Arg Phe Leu Glu Ser Asn Pro Val
1640 1645 1650
Met Ile Asp Ala Lys Glu Val Ser Ala Ala His Arg Ala Arg Tyr
1655 1660 1665
Phe Trp Gly Asn Leu Pro Gly Met Asn Arg Pro Leu Ala Ser Thr
1670 1675 1680
Val Asn Asp Lys Leu Glu Leu Gln Glu Cys Leu Glu His Gly Arg
1685 1690 1695
Ile Ala Lys Phe Ser Lys Val Arg Thr Ile Thr Thr Arg Ser Asn
1700 1705 1710
Ser Ile Lys Gln Gly Lys Asp Gln His Phe Pro Val Phe Met Asn
1715 1720 1725
Glu Lys Glu Asp Ile Leu Trp Cys Thr Glu Met Glu Arg Val Phe
1730 1735 1740
Gly Phe Pro Val His Tyr Thr Asp Val Ser Asn Met Ser Arg Leu
1745 1750 1755
Ala Arg Gln Arg Leu Leu Gly Arg Ser Trp Ser Val Pro Val Ile
1760 1765 1770
Arg His Leu Phe Ala Pro Leu Lys Glu Tyr Phe Ala Cys Val Ser
1775 1780 1785
Ser Gly Asn Ser Asn Ala Asn Ser Arg Gly Pro Ser Phe Ser Ser
1790 1795 1800
Gly Leu Val Pro Leu Ser Leu Arg Gly Ser His Met Gly Pro Met
1805 1810 1815
Glu Ile Tyr Lys Thr Val Ser Ala Trp Lys Arg Gln Pro Val Arg
1820 1825 1830
Val Leu Ser Leu Phe Arg Asn Ile Asp Lys Val Leu Lys Ser Leu
1835 1840 1845
Gly Phe Leu Glu Ser Gly Ser Gly Ser Gly Gly Gly Thr Leu Lys
1850 1855 1860
Tyr Val Glu Asp Val Thr Asn Val Val Arg Arg Asp Val Glu Lys
1865 1870 1875
Trp Gly Pro Phe Asp Leu Val Tyr Gly Ser Thr Gln Pro Leu Gly
1880 1885 1890
Ser Ser Cys Asp Arg Cys Pro Gly Trp Tyr Met Phe Gln Phe His
1895 1900 1905
Arg Ile Leu Gln Tyr Ala Leu Pro Arg Gln Glu Ser Gln Arg Pro
1910 1915 1920
Phe Phe Trp Ile Phe Met Asp Asn Leu Leu Leu Thr Glu Asp Asp
1925 1930 1935
Gln Glu Thr Thr Thr Arg Phe Leu Gln Thr Glu Ala Val Thr Leu
1940 1945 1950
Gln Asp Val Arg Gly Arg Asp Tyr Gln Asn Ala Met Arg Val Trp
1955 1960 1965
Ser Asn Ile Pro Gly Leu Lys Ser Lys His Ala Pro Leu Thr Pro
1970 1975 1980
Lys Glu Glu Glu Tyr Leu Gln Ala Gln Val Arg Ser Arg Ser Lys
1985 1990 1995
Leu Asp Ala Pro Lys Val Asp Leu Leu Val Lys Asn Cys Leu Leu
2000 2005 2010
Pro Leu Arg Glu Tyr Phe Lys Tyr Phe Ser Gln Asn Ser Leu Pro
2015 2020 2025
Leu Ser Arg Ala Asp Pro Lys Lys Lys Arg Lys Val Gly Ser Gly
2030 2035 2040
Ala Thr Asn Phe Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu
2045 2050 2055
Asn Pro Gly Pro Ser Glu Leu Ile Lys Glu Asn Met His Met Lys
2060 2065 2070
Leu Tyr Met Glu Gly Thr Val Asp Asn His His Phe Lys Cys Thr
2075 2080 2085
Ser Glu Gly Glu Gly Lys Pro Tyr Glu Gly Thr Gln Thr Met Arg
2090 2095 2100
Ile Lys Val Val Glu Gly Gly Pro Leu Pro Phe Ala Phe Asp Ile
2105 2110 2115
Leu Ala Thr Ser Phe Leu Tyr Gly Ser Lys Thr Phe Ile Asn His
2120 2125 2130
Thr Gln Gly Ile Pro Asp Phe Phe Lys Gln Ser Phe Pro Glu Gly
2135 2140 2145
Phe Thr Trp Glu Arg Val Thr Thr Tyr Glu Asp Gly Gly Val Leu
2150 2155 2160
Thr Ala Thr Gln Asp Thr Ser Leu Gln Asp Gly Cys Leu Ile Tyr
2165 2170 2175
Asn Val Lys Ile Arg Gly Val Asn Phe Thr Ser Asn Gly Pro Val
2180 2185 2190
Met Gln Lys Lys Thr Leu Gly Trp Glu Ala Phe Thr Glu Thr Leu
2195 2200 2205
Tyr Pro Ala Asp Gly Gly Leu Glu Gly Arg Asn Asp Met Ala Leu
2210 2215 2220
Lys Leu Val Gly Gly Ser His Leu Ile Ala Asn Ile Lys Thr Thr
2225 2230 2235
Tyr Arg Ser Lys Lys Pro Ala Lys Asn Leu Lys Met Pro Gly Val
2240 2245 2250
Tyr Tyr Val Asp Tyr Arg Leu Glu Arg Ile Lys Glu Ala Asn Asn
2255 2260 2265
Glu Thr Tyr Val Glu Gln His Glu Val Ala Val Ala Arg Tyr Cys
2270 2275 2280
Asp Leu Pro Ser Lys Leu Gly His Lys Leu Asn
2285 2290
<210> 2
<211> 2296
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетический полипептид
<400> 2
Asp Ala Lys Ser Leu Thr Ala Trp Ser Arg Thr Leu Val Thr Phe Lys
1. 5 10 15
Asp Val Phe Val Asp Phe Thr Arg Glu Glu Trp Lys Leu Leu Asp Thr
20 25 30
Ala Gln Gln Ile Val Tyr Arg Asn Val Met Leu Glu Asn Tyr Lys Asn
35 40 45
Leu Val Ser Leu Gly Tyr Gln Leu Thr Lys Pro Asp Val Ile Leu Arg
50 55 60
Leu Glu Lys Gly Glu Glu Pro Gly Gly Ser Gly Gly Gly Ser Met Asp
65 70 75 80
Lys Lys Tyr Ser Ile Gly Leu Ala Ile Gly Thr Asn Ser Val Gly Trp
85 90 95
Ala Val Ile Thr Asp Glu Tyr Lys Val Pro Ser Lys Lys Phe Lys Val
100 105 110
Leu Gly Asn Thr Asp Arg His Ser Ile Lys Lys Asn Leu Ile Gly Ala
115 120 125
Leu Leu Phe Asp Ser Gly Glu Thr Ala Glu Ala Thr Arg Leu Lys Arg
130 135 140
Thr Ala Arg Arg Arg Tyr Thr Arg Arg Lys Asn Arg Ile Cys Tyr Leu
145 150 155 160
Gln Glu Ile Phe Ser Asn Glu Met Ala Lys Val Asp Asp Ser Phe Phe
165 170 175
His Arg Leu Glu Glu Ser Phe Leu Val Glu Glu Asp Lys Lys His Glu
180 185 190
Arg His Pro Ile Phe Gly Asn Ile Val Asp Glu Val Ala Tyr His Glu
195 200 205
Lys Tyr Pro Thr Ile Tyr His Leu Arg Lys Lys Leu Val Asp Ser Thr
210 215 220
Asp Lys Ala Asp Leu Arg Leu Ile Tyr Leu Ala Leu Ala His Met Ile
225 230 235 240
Lys Phe Arg Gly His Phe Leu Ile Glu Gly Asp Leu Asn Pro Asp Asn
245 250 255
Ser Asp Val Asp Lys Leu Phe Ile Gln Leu Val Gln Thr Tyr Asn Gln
260 265 270
Leu Phe Glu Glu Asn Pro Ile Asn Ala Ser Gly Val Asp Ala Lys Ala
275 280 285
Ile Leu Ser Ala Arg Leu Ser Lys Ser Arg Arg Leu Glu Asn Leu Ile
290 295 300
Ala Gln Leu Pro Gly Glu Lys Lys Asn Gly Leu Phe Gly Asn Leu Ile
305 310 315 320
Ala Leu Ser Leu Gly Leu Thr Pro Asn Phe Lys Ser Asn Phe Asp Leu
325 330 335
Ala Glu Asp Ala Lys Leu Gln Leu Ser Lys Asp Thr Tyr Asp Asp Asp
340 345 350
Leu Asp Asn Leu Leu Ala Gln Ile Gly Asp Gln Tyr Ala Asp Leu Phe
355 360 365
Leu Ala Ala Lys Asn Leu Ser Asp Ala Ile Leu Leu Ser Asp Ile Leu
370 375 380
Arg Val Asn Thr Glu Ile Thr Lys Ala Pro Leu Ser Ala Ser Met Ile
385 390 395 400
Lys Arg Tyr Asp Glu His His Gln Asp Leu Thr Leu Leu Lys Ala Leu
405 410 415
Val Arg Gln Gln Leu Pro Glu Lys Tyr Lys Glu Ile Phe Phe Asp Gln
420 425 430
Ser Lys Asn Gly Tyr Ala Gly Tyr Ile Asp Gly Gly Ala Ser Gln Glu
435 440 445
Glu Phe Tyr Lys Phe Ile Lys Pro Ile Leu Glu Lys Met Asp Gly Thr
450 455 460
Glu Glu Leu Leu Val Lys Leu Asn Arg Glu Asp Leu Leu Arg Lys Gln
465 470 475 480
Arg Thr Phe Asp Asn Gly Ser Ile Pro His Gln Ile His Leu Gly Glu
485 490 495
Leu His Ala Ile Leu Arg Arg Gln Glu Asp Phe Tyr Pro Phe Leu Lys
500 505 510
Asp Asn Arg Glu Lys Ile Glu Lys Ile Leu Thr Phe Arg Ile Pro Tyr
515 520 525
Tyr Val Gly Pro Leu Ala Arg Gly Asn Ser Arg Phe Ala Trp Met Thr
530 535 540
Arg Lys Ser Glu Glu Thr Ile Thr Pro Trp Asn Phe Glu Glu Val Val
545 550 555 560
Asp Lys Gly Ala Ser Ala Gln Ser Phe Ile Glu Arg Met Thr Asn Phe
565 570 575
Asp Lys Asn Leu Pro Asn Glu Lys Val Leu Pro Lys His Ser Leu Leu
580 585 590
Tyr Glu Tyr Phe Thr Val Tyr Asn Glu Leu Thr Lys Val Lys Tyr Val
595 600 605
Thr Glu Gly Met Arg Lys Pro Ala Phe Leu Ser Gly Glu Gln Lys Lys
610 615 620
Ala Ile Val Asp Leu Leu Phe Lys Thr Asn Arg Lys Val Thr Val Lys
625 630 635 640
Gln Leu Lys Glu Asp Tyr Phe Lys Lys Ile Glu Cys Phe Asp Ser Val
645 650 655
Glu Ile Ser Gly Val Glu Asp Arg Phe Asn Ala Ser Leu Gly Thr Tyr
660 665 670
His Asp Leu Leu Lys Ile Ile Lys Asp Lys Asp Phe Leu Asp Asn Glu
675 680 685
Glu Asn Glu Asp Ile Leu Glu Asp Ile Val Leu Thr Leu Thr Leu Phe
690 695 700
Glu Asp Arg Glu Met Ile Glu Glu Arg Leu Lys Thr Tyr Ala His Leu
705 710 715 720
Phe Asp Asp Lys Val Met Lys Gln Leu Lys Arg Arg Arg Tyr Thr Gly
725 730 735
Trp Gly Arg Leu Ser Arg Lys Leu Ile Asn Gly Ile Arg Asp Lys Gln
740 745 750
Ser Gly Lys Thr Ile Leu Asp Phe Leu Lys Ser Asp Gly Phe Ala Asn
755 760 765
Arg Asn Phe Met Gln Leu Ile His Asp Asp Ser Leu Thr Phe Lys Glu
770 775 780
Asp Ile Gln Lys Ala Gln Val Ser Gly Gln Gly Asp Ser Leu His Glu
785 790 795 800
His Ile Ala Asn Leu Ala Gly Ser Pro Ala Ile Lys Lys Gly Ile Leu
805 810 815
Gln Thr Val Lys Val Val Asp Glu Leu Val Lys Val Met Gly Arg His
820 825 830
Lys Pro Glu Asn Ile Val Ile Glu Met Ala Arg Glu Asn Gln Thr Thr
835 840 845
Gln Lys Gly Gln Lys Asn Ser Arg Glu Arg Met Lys Arg Ile Glu Glu
850 855 860
Gly Ile Lys Glu Leu Gly Ser Gln Ile Leu Lys Glu His Pro Val Glu
865 870 875 880
Asn Thr Gln Leu Gln Asn Glu Lys Leu Tyr Leu Tyr Tyr Leu Gln Asn
885 890 895
Gly Arg Asp Met Tyr Val Asp Gln Glu Leu Asp Ile Asn Arg Leu Ser
900 905 910
Asp Tyr Asp Val Asp Ala Ile Val Pro Gln Ser Phe Leu Lys Asp Asp
915 920 925
Ser Ile Asp Asn Lys Val Leu Thr Arg Ser Asp Lys Asn Arg Gly Lys
930 935 940
Ser Asp Asn Val Pro Ser Glu Glu Val Val Lys Lys Met Lys Asn Tyr
945 950 955 960
Trp Arg Gln Leu Leu Asn Ala Lys Leu Ile Thr Gln Arg Lys Phe Asp
965 970 975
Asn Leu Thr Lys Ala Glu Arg Gly Gly Leu Ser Glu Leu Asp Lys Ala
980 985 990
Gly Phe Ile Lys Arg Gln Leu Val Glu Thr Arg Gln Ile Thr Lys His
995 1000 1005
Val Ala Gln Ile Leu Asp Ser Arg Met Asn Thr Lys Tyr Asp Glu
1010 1015 1020
Asn Asp Lys Leu Ile Arg Glu Val Lys Val Ile Thr Leu Lys Ser
1025 1030 1035
Lys Leu Val Ser Asp Phe Arg Lys Asp Phe Gln Phe Tyr Lys Val
1040 1045 1050
Arg Glu Ile Asn Asn Tyr His His Ala His Asp Ala Tyr Leu Asn
1055 1060 1065
Ala Val Val Gly Thr Ala Leu Ile Lys Lys Tyr Pro Lys Leu Glu
1070 1075 1080
Ser Glu Phe Val Tyr Gly Asp Tyr Lys Val Tyr Asp Val Arg Lys
1085 1090 1095
Met Ile Ala Lys Ser Glu Gln Glu Ile Gly Lys Ala Thr Ala Lys
1100 1105 1110
Tyr Phe Phe Tyr Ser Asn Ile Met Asn Phe Phe Lys Thr Glu Ile
1115 1120 1125
Thr Leu Ala Asn Gly Glu Ile Arg Lys Arg Pro Leu Ile Glu Thr
1130 1135 1140
Asn Gly Glu Thr Gly Glu Ile Val Trp Asp Lys Gly Arg Asp Phe
1145 1150 1155
Ala Thr Val Arg Lys Val Leu Ser Met Pro Gln Val Asn Ile Val
1160 1165 1170
Lys Lys Thr Glu Val Gln Thr Gly Gly Phe Ser Lys Glu Ser Ile
1175 1180 1185
Leu Pro Lys Arg Asn Ser Asp Lys Leu Ile Ala Arg Lys Lys Asp
1190 1195 1200
Trp Asp Pro Lys Lys Tyr Gly Gly Phe Asp Ser Pro Thr Val Ala
1205 1210 1215
Tyr Ser Val Leu Val Val Ala Lys Val Glu Lys Gly Lys Ser Lys
1220 1225 1230
Lys Leu Lys Ser Val Lys Glu Leu Leu Gly Ile Thr Ile Met Glu
1235 1240 1245
Arg Ser Ser Phe Glu Lys Asn Pro Ile Asp Phe Leu Glu Ala Lys
1250 1255 1260
Gly Tyr Lys Glu Val Lys Lys Asp Leu Ile Ile Lys Leu Pro Lys
1265 1270 1275
Tyr Ser Leu Phe Glu Leu Glu Asn Gly Arg Lys Arg Met Leu Ala
1280 1285 1290
Ser Ala Gly Glu Leu Gln Lys Gly Asn Glu Leu Ala Leu Pro Ser
1295 1300 1305
Lys Tyr Val Asn Phe Leu Tyr Leu Ala Ser His Tyr Glu Lys Leu
1310 1315 1320
Lys Gly Ser Pro Glu Asp Asn Glu Gln Lys Gln Leu Phe Val Glu
1325 1330 1335
Gln His Lys His Tyr Leu Asp Glu Ile Ile Glu Gln Ile Ser Glu
1340 1345 1350
Phe Ser Lys Arg Val Ile Leu Ala Asp Ala Asn Leu Asp Lys Val
1355 1360 1365
Leu Ser Ala Tyr Asn Lys His Arg Asp Lys Pro Ile Arg Glu Gln
1370 1375 1380
Ala Glu Asn Ile Ile His Leu Phe Thr Leu Thr Asn Leu Gly Ala
1385 1390 1395
Pro Ala Ala Phe Lys Tyr Phe Asp Thr Thr Ile Asp Arg Lys Arg
1400 1405 1410
Tyr Thr Ser Thr Lys Glu Val Leu Asp Ala Thr Leu Ile His Gln
1415 1420 1425
Ser Ile Thr Gly Leu Tyr Glu Thr Arg Ile Asp Leu Ser Gln Leu
1430 1435 1440
Gly Gly Asp Ser Arg Ala Asp Tyr Pro Tyr Asp Val Pro Asp Tyr
1445 1450 1455
Ala Ser Gly Ser Pro Lys Lys Lys Arg Lys Val Ser Pro Gly Ser
1460 1465 1470
Gly Ser Glu Thr Pro Gly Thr Ser Glu Ser Ala Thr Pro Glu Ser
1475 1480 1485
Asn His Asp Gln Glu Phe Asp Pro Pro Lys Val Tyr Pro Pro Val
1490 1495 1500
Pro Ala Glu Lys Arg Lys Pro Ile Arg Val Leu Ser Leu Phe Asp
1505 1510 1515
Gly Ile Ala Thr Gly Leu Leu Val Leu Lys Asp Leu Gly Ile Gln
1520 1525 1530
Val Asp Arg Tyr Ile Ala Ser Glu Val Cys Glu Asp Ser Ile Thr
1535 1540 1545
Val Gly Met Val Arg His Gln Gly Lys Ile Met Tyr Val Gly Asp
1550 1555 1560
Val Arg Ser Val Thr Gln Lys His Ile Gln Glu Trp Gly Pro Phe
1565 1570 1575
Asp Leu Val Ile Gly Gly Ser Pro Cys Asn Asp Leu Ser Ile Val
1580 1585 1590
Asn Pro Ala Arg Lys Gly Leu Tyr Glu Gly Thr Gly Arg Leu Phe
1595 1600 1605
Phe Glu Phe Tyr Arg Leu Leu His Asp Ala Arg Pro Lys Glu Gly
1610 1615 1620
Asp Asp Arg Pro Phe Phe Trp Leu Phe Glu Asn Val Val Ala Met
1625 1630 1635
Gly Val Ser Asp Lys Arg Asp Ile Ser Arg Phe Leu Glu Ser Asn
1640 1645 1650
Pro Val Met Ile Asp Ala Lys Glu Val Ser Ala Ala His Arg Ala
1655 1660 1665
Arg Tyr Phe Trp Gly Asn Leu Pro Gly Met Asn Arg Pro Leu Ala
1670 1675 1680
Ser Thr Val Asn Asp Lys Leu Glu Leu Gln Glu Cys Leu Glu His
1685 1690 1695
Gly Arg Ile Ala Lys Phe Ser Lys Val Arg Thr Ile Thr Thr Arg
1700 1705 1710
Ser Asn Ser Ile Lys Gln Gly Lys Asp Gln His Phe Pro Val Phe
1715 1720 1725
Met Asn Glu Lys Glu Asp Ile Leu Trp Cys Thr Glu Met Glu Arg
1730 1735 1740
Val Phe Gly Phe Pro Val His Tyr Thr Asp Val Ser Asn Met Ser
1745 1750 1755
Arg Leu Ala Arg Gln Arg Leu Leu Gly Arg Ser Trp Ser Val Pro
1760 1765 1770
Val Ile Arg His Leu Phe Ala Pro Leu Lys Glu Tyr Phe Ala Cys
1775 1780 1785
Val Ser Ser Gly Asn Ser Asn Ala Asn Ser Arg Gly Pro Ser Phe
1790 1795 1800
Ser Ser Gly Leu Val Pro Leu Ser Leu Arg Gly Ser His Met Gly
1805 1810 1815
Pro Met Glu Ile Tyr Lys Thr Val Ser Ala Trp Lys Arg Gln Pro
1820 1825 1830
Val Arg Val Leu Ser Leu Phe Arg Asn Ile Asp Lys Val Leu Lys
1835 1840 1845
Ser Leu Gly Phe Leu Glu Ser Gly Ser Gly Ser Gly Gly Gly Thr
1850 1855 1860
Leu Lys Tyr Val Glu Asp Val Thr Asn Val Val Arg Arg Asp Val
1865 1870 1875
Glu Lys Trp Gly Pro Phe Asp Leu Val Tyr Gly Ser Thr Gln Pro
1880 1885 1890
Leu Gly Ser Ser Cys Asp Arg Cys Pro Gly Trp Tyr Met Phe Gln
1895 1900 1905
Phe His Arg Ile Leu Gln Tyr Ala Leu Pro Arg Gln Glu Ser Gln
1910 1915 1920
Arg Pro Phe Phe Trp Ile Phe Met Asp Asn Leu Leu Leu Thr Glu
1925 1930 1935
Asp Asp Gln Glu Thr Thr Thr Arg Phe Leu Gln Thr Glu Ala Val
1940 1945 1950
Thr Leu Gln Asp Val Arg Gly Arg Asp Tyr Gln Asn Ala Met Arg
1955 1960 1965
Val Trp Ser Asn Ile Pro Gly Leu Lys Ser Lys His Ala Pro Leu
1970 1975 1980
Thr Pro Lys Glu Glu Glu Tyr Leu Gln Ala Gln Val Arg Ser Arg
1985 1990 1995
Ser Lys Leu Asp Ala Pro Lys Val Asp Leu Leu Val Lys Asn Cys
2000 2005 2010
Leu Leu Pro Leu Arg Glu Tyr Phe Lys Tyr Phe Ser Gln Asn Ser
2015 2020 2025
Leu Pro Leu Ser Arg Ala Asp Pro Lys Lys Lys Arg Lys Val Gly
2030 2035 2040
Ser Gly Ala Thr Asn Phe Ser Leu Leu Lys Gln Ala Gly Asp Val
2045 2050 2055
Glu Glu Asn Pro Gly Pro Ser Glu Leu Ile Lys Glu Asn Met His
2060 2065 2070
Met Lys Leu Tyr Met Glu Gly Thr Val Asp Asn His His Phe Lys
2075 2080 2085
Cys Thr Ser Glu Gly Glu Gly Lys Pro Tyr Glu Gly Thr Gln Thr
2090 2095 2100
Met Arg Ile Lys Val Val Glu Gly Gly Pro Leu Pro Phe Ala Phe
2105 2110 2115
Asp Ile Leu Ala Thr Ser Phe Leu Tyr Gly Ser Lys Thr Phe Ile
2120 2125 2130
Asn His Thr Gln Gly Ile Pro Asp Phe Phe Lys Gln Ser Phe Pro
2135 2140 2145
Glu Gly Phe Thr Trp Glu Arg Val Thr Thr Tyr Glu Asp Gly Gly
2150 2155 2160
Val Leu Thr Ala Thr Gln Asp Thr Ser Leu Gln Asp Gly Cys Leu
2165 2170 2175
Ile Tyr Asn Val Lys Ile Arg Gly Val Asn Phe Thr Ser Asn Gly
2180 2185 2190
Pro Val Met Gln Lys Lys Thr Leu Gly Trp Glu Ala Phe Thr Glu
2195 2200 2205
Thr Leu Tyr Pro Ala Asp Gly Gly Leu Glu Gly Arg Asn Asp Met
2210 2215 2220
Ala Leu Lys Leu Val Gly Gly Ser His Leu Ile Ala Asn Ile Lys
2225 2230 2235
Thr Thr Tyr Arg Ser Lys Lys Pro Ala Lys Asn Leu Lys Met Pro
2240 2245 2250
Gly Val Tyr Tyr Val Asp Tyr Arg Leu Glu Arg Ile Lys Glu Ala
2255 2260 2265
Asn Asn Glu Thr Tyr Val Glu Gln His Glu Val Ala Val Ala Arg
2270 2275 2280
Tyr Cys Asp Leu Pro Ser Lys Leu Gly His Lys Leu Asn
2285 2290 2295
<210> 3
<211> 2316
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетический полипептид
<400> 3
Asp Ala Lys Ser Leu Thr Ala Trp Ser Arg Thr Leu Val Thr Phe Lys
1. 5 10 15
Asp Val Phe Val Asp Phe Thr Arg Glu Glu Trp Lys Leu Leu Asp Thr
20 25 30
Ala Gln Gln Ile Val Tyr Arg Asn Val Met Leu Glu Asn Tyr Lys Asn
35 40 45
Leu Val Ser Leu Gly Tyr Gln Leu Thr Lys Pro Asp Val Ile Leu Arg
50 55 60
Leu Glu Lys Gly Glu Glu Pro Gly Gly Ser Gly Gly Gly Ser Met Asp
65 70 75 80
Lys Lys Tyr Ser Ile Gly Leu Ala Ile Gly Thr Asn Ser Val Gly Trp
85 90 95
Ala Val Ile Thr Asp Glu Tyr Lys Val Pro Ser Lys Lys Phe Lys Val
100 105 110
Leu Gly Asn Thr Asp Arg His Ser Ile Lys Lys Asn Leu Ile Gly Ala
115 120 125
Leu Leu Phe Asp Ser Gly Glu Thr Ala Glu Ala Thr Arg Leu Lys Arg
130 135 140
Thr Ala Arg Arg Arg Tyr Thr Arg Arg Lys Asn Arg Ile Cys Tyr Leu
145 150 155 160
Gln Glu Ile Phe Ser Asn Glu Met Ala Lys Val Asp Asp Ser Phe Phe
165 170 175
His Arg Leu Glu Glu Ser Phe Leu Val Glu Glu Asp Lys Lys His Glu
180 185 190
Arg His Pro Ile Phe Gly Asn Ile Val Asp Glu Val Ala Tyr His Glu
195 200 205
Lys Tyr Pro Thr Ile Tyr His Leu Arg Lys Lys Leu Val Asp Ser Thr
210 215 220
Asp Lys Ala Asp Leu Arg Leu Ile Tyr Leu Ala Leu Ala His Met Ile
225 230 235 240
Lys Phe Arg Gly His Phe Leu Ile Glu Gly Asp Leu Asn Pro Asp Asn
245 250 255
Ser Asp Val Asp Lys Leu Phe Ile Gln Leu Val Gln Thr Tyr Asn Gln
260 265 270
Leu Phe Glu Glu Asn Pro Ile Asn Ala Ser Gly Val Asp Ala Lys Ala
275 280 285
Ile Leu Ser Ala Arg Leu Ser Lys Ser Arg Arg Leu Glu Asn Leu Ile
290 295 300
Ala Gln Leu Pro Gly Glu Lys Lys Asn Gly Leu Phe Gly Asn Leu Ile
305 310 315 320
Ala Leu Ser Leu Gly Leu Thr Pro Asn Phe Lys Ser Asn Phe Asp Leu
325 330 335
Ala Glu Asp Ala Lys Leu Gln Leu Ser Lys Asp Thr Tyr Asp Asp Asp
340 345 350
Leu Asp Asn Leu Leu Ala Gln Ile Gly Asp Gln Tyr Ala Asp Leu Phe
355 360 365
Leu Ala Ala Lys Asn Leu Ser Asp Ala Ile Leu Leu Ser Asp Ile Leu
370 375 380
Arg Val Asn Thr Glu Ile Thr Lys Ala Pro Leu Ser Ala Ser Met Ile
385 390 395 400
Lys Arg Tyr Asp Glu His His Gln Asp Leu Thr Leu Leu Lys Ala Leu
405 410 415
Val Arg Gln Gln Leu Pro Glu Lys Tyr Lys Glu Ile Phe Phe Asp Gln
420 425 430
Ser Lys Asn Gly Tyr Ala Gly Tyr Ile Asp Gly Gly Ala Ser Gln Glu
435 440 445
Glu Phe Tyr Lys Phe Ile Lys Pro Ile Leu Glu Lys Met Asp Gly Thr
450 455 460
Glu Glu Leu Leu Val Lys Leu Asn Arg Glu Asp Leu Leu Arg Lys Gln
465 470 475 480
Arg Thr Phe Asp Asn Gly Ser Ile Pro His Gln Ile His Leu Gly Glu
485 490 495
Leu His Ala Ile Leu Arg Arg Gln Glu Asp Phe Tyr Pro Phe Leu Lys
500 505 510
Asp Asn Arg Glu Lys Ile Glu Lys Ile Leu Thr Phe Arg Ile Pro Tyr
515 520 525
Tyr Val Gly Pro Leu Ala Arg Gly Asn Ser Arg Phe Ala Trp Met Thr
530 535 540
Arg Lys Ser Glu Glu Thr Ile Thr Pro Trp Asn Phe Glu Glu Val Val
545 550 555 560
Asp Lys Gly Ala Ser Ala Gln Ser Phe Ile Glu Arg Met Thr Asn Phe
565 570 575
Asp Lys Asn Leu Pro Asn Glu Lys Val Leu Pro Lys His Ser Leu Leu
580 585 590
Tyr Glu Tyr Phe Thr Val Tyr Asn Glu Leu Thr Lys Val Lys Tyr Val
595 600 605
Thr Glu Gly Met Arg Lys Pro Ala Phe Leu Ser Gly Glu Gln Lys Lys
610 615 620
Ala Ile Val Asp Leu Leu Phe Lys Thr Asn Arg Lys Val Thr Val Lys
625 630 635 640
Gln Leu Lys Glu Asp Tyr Phe Lys Lys Ile Glu Cys Phe Asp Ser Val
645 650 655
Glu Ile Ser Gly Val Glu Asp Arg Phe Asn Ala Ser Leu Gly Thr Tyr
660 665 670
His Asp Leu Leu Lys Ile Ile Lys Asp Lys Asp Phe Leu Asp Asn Glu
675 680 685
Glu Asn Glu Asp Ile Leu Glu Asp Ile Val Leu Thr Leu Thr Leu Phe
690 695 700
Glu Asp Arg Glu Met Ile Glu Glu Arg Leu Lys Thr Tyr Ala His Leu
705 710 715 720
Phe Asp Asp Lys Val Met Lys Gln Leu Lys Arg Arg Arg Tyr Thr Gly
725 730 735
Trp Gly Arg Leu Ser Arg Lys Leu Ile Asn Gly Ile Arg Asp Lys Gln
740 745 750
Ser Gly Lys Thr Ile Leu Asp Phe Leu Lys Ser Asp Gly Phe Ala Asn
755 760 765
Arg Asn Phe Met Gln Leu Ile His Asp Asp Ser Leu Thr Phe Lys Glu
770 775 780
Asp Ile Gln Lys Ala Gln Val Ser Gly Gln Gly Asp Ser Leu His Glu
785 790 795 800
His Ile Ala Asn Leu Ala Gly Ser Pro Ala Ile Lys Lys Gly Ile Leu
805 810 815
Gln Thr Val Lys Val Val Asp Glu Leu Val Lys Val Met Gly Arg His
820 825 830
Lys Pro Glu Asn Ile Val Ile Glu Met Ala Arg Glu Asn Gln Thr Thr
835 840 845
Gln Lys Gly Gln Lys Asn Ser Arg Glu Arg Met Lys Arg Ile Glu Glu
850 855 860
Gly Ile Lys Glu Leu Gly Ser Gln Ile Leu Lys Glu His Pro Val Glu
865 870 875 880
Asn Thr Gln Leu Gln Asn Glu Lys Leu Tyr Leu Tyr Tyr Leu Gln Asn
885 890 895
Gly Arg Asp Met Tyr Val Asp Gln Glu Leu Asp Ile Asn Arg Leu Ser
900 905 910
Asp Tyr Asp Val Asp Ala Ile Val Pro Gln Ser Phe Leu Lys Asp Asp
915 920 925
Ser Ile Asp Asn Lys Val Leu Thr Arg Ser Asp Lys Asn Arg Gly Lys
930 935 940
Ser Asp Asn Val Pro Ser Glu Glu Val Val Lys Lys Met Lys Asn Tyr
945 950 955 960
Trp Arg Gln Leu Leu Asn Ala Lys Leu Ile Thr Gln Arg Lys Phe Asp
965 970 975
Asn Leu Thr Lys Ala Glu Arg Gly Gly Leu Ser Glu Leu Asp Lys Ala
980 985 990
Gly Phe Ile Lys Arg Gln Leu Val Glu Thr Arg Gln Ile Thr Lys His
995 1000 1005
Val Ala Gln Ile Leu Asp Ser Arg Met Asn Thr Lys Tyr Asp Glu
1010 1015 1020
Asn Asp Lys Leu Ile Arg Glu Val Lys Val Ile Thr Leu Lys Ser
1025 1030 1035
Lys Leu Val Ser Asp Phe Arg Lys Asp Phe Gln Phe Tyr Lys Val
1040 1045 1050
Arg Glu Ile Asn Asn Tyr His His Ala His Asp Ala Tyr Leu Asn
1055 1060 1065
Ala Val Val Gly Thr Ala Leu Ile Lys Lys Tyr Pro Lys Leu Glu
1070 1075 1080
Ser Glu Phe Val Tyr Gly Asp Tyr Lys Val Tyr Asp Val Arg Lys
1085 1090 1095
Met Ile Ala Lys Ser Glu Gln Glu Ile Gly Lys Ala Thr Ala Lys
1100 1105 1110
Tyr Phe Phe Tyr Ser Asn Ile Met Asn Phe Phe Lys Thr Glu Ile
1115 1120 1125
Thr Leu Ala Asn Gly Glu Ile Arg Lys Arg Pro Leu Ile Glu Thr
1130 1135 1140
Asn Gly Glu Thr Gly Glu Ile Val Trp Asp Lys Gly Arg Asp Phe
1145 1150 1155
Ala Thr Val Arg Lys Val Leu Ser Met Pro Gln Val Asn Ile Val
1160 1165 1170
Lys Lys Thr Glu Val Gln Thr Gly Gly Phe Ser Lys Glu Ser Ile
1175 1180 1185
Leu Pro Lys Arg Asn Ser Asp Lys Leu Ile Ala Arg Lys Lys Asp
1190 1195 1200
Trp Asp Pro Lys Lys Tyr Gly Gly Phe Asp Ser Pro Thr Val Ala
1205 1210 1215
Tyr Ser Val Leu Val Val Ala Lys Val Glu Lys Gly Lys Ser Lys
1220 1225 1230
Lys Leu Lys Ser Val Lys Glu Leu Leu Gly Ile Thr Ile Met Glu
1235 1240 1245
Arg Ser Ser Phe Glu Lys Asn Pro Ile Asp Phe Leu Glu Ala Lys
1250 1255 1260
Gly Tyr Lys Glu Val Lys Lys Asp Leu Ile Ile Lys Leu Pro Lys
1265 1270 1275
Tyr Ser Leu Phe Glu Leu Glu Asn Gly Arg Lys Arg Met Leu Ala
1280 1285 1290
Ser Ala Gly Glu Leu Gln Lys Gly Asn Glu Leu Ala Leu Pro Ser
1295 1300 1305
Lys Tyr Val Asn Phe Leu Tyr Leu Ala Ser His Tyr Glu Lys Leu
1310 1315 1320
Lys Gly Ser Pro Glu Asp Asn Glu Gln Lys Gln Leu Phe Val Glu
1325 1330 1335
Gln His Lys His Tyr Leu Asp Glu Ile Ile Glu Gln Ile Ser Glu
1340 1345 1350
Phe Ser Lys Arg Val Ile Leu Ala Asp Ala Asn Leu Asp Lys Val
1355 1360 1365
Leu Ser Ala Tyr Asn Lys His Arg Asp Lys Pro Ile Arg Glu Gln
1370 1375 1380
Ala Glu Asn Ile Ile His Leu Phe Thr Leu Thr Asn Leu Gly Ala
1385 1390 1395
Pro Ala Ala Phe Lys Tyr Phe Asp Thr Thr Ile Asp Arg Lys Arg
1400 1405 1410
Tyr Thr Ser Thr Lys Glu Val Leu Asp Ala Thr Leu Ile His Gln
1415 1420 1425
Ser Ile Thr Gly Leu Tyr Glu Thr Arg Ile Asp Leu Ser Gln Leu
1430 1435 1440
Gly Gly Asp Ser Arg Ala Asp Tyr Pro Tyr Asp Val Pro Asp Tyr
1445 1450 1455
Ala Ser Gly Ser Pro Lys Lys Lys Arg Lys Val Glu Ala Ser Gly
1460 1465 1470
Ser Gly Arg Ala Ser Pro Gly Ile Pro Gly Ser Thr Arg Asn His
1475 1480 1485
Asp Gln Glu Phe Asp Pro Pro Lys Val Tyr Pro Pro Val Pro Ala
1490 1495 1500
Glu Lys Arg Lys Pro Ile Arg Val Leu Ser Leu Phe Asp Gly Ile
1505 1510 1515
Ala Thr Gly Leu Leu Val Leu Lys Asp Leu Gly Ile Gln Val Asp
1520 1525 1530
Arg Tyr Ile Ala Ser Glu Val Cys Glu Asp Ser Ile Thr Val Gly
1535 1540 1545
Met Val Arg His Gln Gly Lys Ile Met Tyr Val Gly Asp Val Arg
1550 1555 1560
Ser Val Thr Gln Lys His Ile Gln Glu Trp Gly Pro Phe Asp Leu
1565 1570 1575
Val Ile Gly Gly Ser Pro Cys Asn Asp Leu Ser Ile Val Asn Pro
1580 1585 1590
Ala Arg Lys Gly Leu Tyr Glu Gly Thr Gly Arg Leu Phe Phe Glu
1595 1600 1605
Phe Tyr Arg Leu Leu His Asp Ala Arg Pro Lys Glu Gly Asp Asp
1610 1615 1620
Arg Pro Phe Phe Trp Leu Phe Glu Asn Val Val Ala Met Gly Val
1625 1630 1635
Ser Asp Lys Arg Asp Ile Ser Arg Phe Leu Glu Ser Asn Pro Val
1640 1645 1650
Met Ile Asp Ala Lys Glu Val Ser Ala Ala His Arg Ala Arg Tyr
1655 1660 1665
Phe Trp Gly Asn Leu Pro Gly Met Asn Arg Pro Leu Ala Ser Thr
1670 1675 1680
Val Asn Asp Lys Leu Glu Leu Gln Glu Cys Leu Glu His Gly Arg
1685 1690 1695
Ile Ala Lys Phe Ser Lys Val Arg Thr Ile Thr Thr Arg Ser Asn
1700 1705 1710
Ser Ile Lys Gln Gly Lys Asp Gln His Phe Pro Val Phe Met Asn
1715 1720 1725
Glu Lys Glu Asp Ile Leu Trp Cys Thr Glu Met Glu Arg Val Phe
1730 1735 1740
Gly Phe Pro Val His Tyr Thr Asp Val Ser Asn Met Ser Arg Leu
1745 1750 1755
Ala Arg Gln Arg Leu Leu Gly Arg Ser Trp Ser Val Pro Val Ile
1760 1765 1770
Arg His Leu Phe Ala Pro Leu Lys Glu Tyr Phe Ala Cys Val Ser
1775 1780 1785
Ser Gly Asn Ser Asn Ala Asn Ser Arg Gly Pro Ser Phe Ser Ser
1790 1795 1800
Gly Leu Val Pro Leu Ser Leu Arg Gly Ser His Met Gly Pro Met
1805 1810 1815
Glu Ile Tyr Lys Thr Val Ser Ala Trp Lys Arg Gln Pro Val Arg
1820 1825 1830
Val Leu Ser Leu Phe Arg Asn Ile Asp Lys Val Leu Lys Ser Leu
1835 1840 1845
Gly Phe Leu Glu Ser Gly Ser Gly Ser Gly Gly Gly Thr Leu Lys
1850 1855 1860
Tyr Val Glu Asp Val Thr Asn Val Val Arg Arg Asp Val Glu Lys
1865 1870 1875
Trp Gly Pro Phe Asp Leu Val Tyr Gly Ser Thr Gln Pro Leu Gly
1880 1885 1890
Ser Ser Cys Asp Arg Cys Pro Gly Trp Tyr Met Phe Gln Phe His
1895 1900 1905
Arg Ile Leu Gln Tyr Ala Leu Pro Arg Gln Glu Ser Gln Arg Pro
1910 1915 1920
Phe Phe Trp Ile Phe Met Asp Asn Leu Leu Leu Thr Glu Asp Asp
1925 1930 1935
Gln Glu Thr Thr Thr Arg Phe Leu Gln Thr Glu Ala Val Thr Leu
1940 1945 1950
Gln Asp Val Arg Gly Arg Asp Tyr Gln Asn Ala Met Arg Val Trp
1955 1960 1965
Ser Asn Ile Pro Gly Leu Lys Ser Lys His Ala Pro Leu Thr Pro
1970 1975 1980
Lys Glu Glu Glu Tyr Leu Gln Ala Gln Val Arg Ser Arg Ser Lys
1985 1990 1995
Leu Asp Ala Pro Lys Val Asp Leu Leu Val Lys Asn Cys Leu Leu
2000 2005 2010
Pro Leu Arg Glu Tyr Phe Lys Tyr Phe Ser Gln Asn Ser Leu Pro
2015 2020 2025
Leu Ser Arg Ala Asp Pro Lys Lys Lys Arg Lys Val Gly Ser Gly
2030 2035 2040
Ala Thr Asn Phe Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu
2045 2050 2055
Asn Pro Gly Pro Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu Lys
2060 2065 2070
Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Ser Glu Leu Ile
2075 2080 2085
Lys Glu Asn Met His Met Lys Leu Tyr Met Glu Gly Thr Val Asp
2090 2095 2100
Asn His His Phe Lys Cys Thr Ser Glu Gly Glu Gly Lys Pro Tyr
2105 2110 2115
Glu Gly Thr Gln Thr Met Arg Ile Lys Val Val Glu Gly Gly Pro
2120 2125 2130
Leu Pro Phe Ala Phe Asp Ile Leu Ala Thr Ser Phe Leu Tyr Gly
2135 2140 2145
Ser Lys Thr Phe Ile Asn His Thr Gln Gly Ile Pro Asp Phe Phe
2150 2155 2160
Lys Gln Ser Phe Pro Glu Gly Phe Thr Trp Glu Arg Val Thr Thr
2165 2170 2175
Tyr Glu Asp Gly Gly Val Leu Thr Ala Thr Gln Asp Thr Ser Leu
2180 2185 2190
Gln Asp Gly Cys Leu Ile Tyr Asn Val Lys Ile Arg Gly Val Asn
2195 2200 2205
Phe Thr Ser Asn Gly Pro Val Met Gln Lys Lys Thr Leu Gly Trp
2210 2215 2220
Glu Ala Phe Thr Glu Thr Leu Tyr Pro Ala Asp Gly Gly Leu Glu
2225 2230 2235
Gly Arg Asn Asp Met Ala Leu Lys Leu Val Gly Gly Ser His Leu
2240 2245 2250
Ile Ala Asn Ile Lys Thr Thr Tyr Arg Ser Lys Lys Pro Ala Lys
2255 2260 2265
Asn Leu Lys Met Pro Gly Val Tyr Tyr Val Asp Tyr Arg Leu Glu
2270 2275 2280
Arg Ile Lys Glu Ala Asn Asn Glu Thr Tyr Val Glu Gln His Glu
2285 2290 2295
Val Ala Val Ala Arg Tyr Cys Asp Leu Pro Ser Lys Leu Gly His
2300 2305 2310
Lys Leu Asn
2315
<210> 4
<211> 2318
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетический полипептид
<400> 4
Asp Ala Lys Ser Leu Thr Ala Trp Ser Arg Thr Leu Val Thr Phe Lys
1. 5 10 15
Asp Val Phe Val Asp Phe Thr Arg Glu Glu Trp Lys Leu Leu Asp Thr
20 25 30
Ala Gln Gln Ile Val Tyr Arg Asn Val Met Leu Glu Asn Tyr Lys Asn
35 40 45
Leu Val Ser Leu Gly Tyr Gln Leu Thr Lys Pro Asp Val Ile Leu Arg
50 55 60
Leu Glu Lys Gly Glu Glu Pro Gly Gly Ser Gly Gly Gly Ser Met Asp
65 70 75 80
Lys Lys Tyr Ser Ile Gly Leu Ala Ile Gly Thr Asn Ser Val Gly Trp
85 90 95
Ala Val Ile Thr Asp Glu Tyr Lys Val Pro Ser Lys Lys Phe Lys Val
100 105 110
Leu Gly Asn Thr Asp Arg His Ser Ile Lys Lys Asn Leu Ile Gly Ala
115 120 125
Leu Leu Phe Asp Ser Gly Glu Thr Ala Glu Ala Thr Arg Leu Lys Arg
130 135 140
Thr Ala Arg Arg Arg Tyr Thr Arg Arg Lys Asn Arg Ile Cys Tyr Leu
145 150 155 160
Gln Glu Ile Phe Ser Asn Glu Met Ala Lys Val Asp Asp Ser Phe Phe
165 170 175
His Arg Leu Glu Glu Ser Phe Leu Val Glu Glu Asp Lys Lys His Glu
180 185 190
Arg His Pro Ile Phe Gly Asn Ile Val Asp Glu Val Ala Tyr His Glu
195 200 205
Lys Tyr Pro Thr Ile Tyr His Leu Arg Lys Lys Leu Val Asp Ser Thr
210 215 220
Asp Lys Ala Asp Leu Arg Leu Ile Tyr Leu Ala Leu Ala His Met Ile
225 230 235 240
Lys Phe Arg Gly His Phe Leu Ile Glu Gly Asp Leu Asn Pro Asp Asn
245 250 255
Ser Asp Val Asp Lys Leu Phe Ile Gln Leu Val Gln Thr Tyr Asn Gln
260 265 270
Leu Phe Glu Glu Asn Pro Ile Asn Ala Ser Gly Val Asp Ala Lys Ala
275 280 285
Ile Leu Ser Ala Arg Leu Ser Lys Ser Arg Arg Leu Glu Asn Leu Ile
290 295 300
Ala Gln Leu Pro Gly Glu Lys Lys Asn Gly Leu Phe Gly Asn Leu Ile
305 310 315 320
Ala Leu Ser Leu Gly Leu Thr Pro Asn Phe Lys Ser Asn Phe Asp Leu
325 330 335
Ala Glu Asp Ala Lys Leu Gln Leu Ser Lys Asp Thr Tyr Asp Asp Asp
340 345 350
Leu Asp Asn Leu Leu Ala Gln Ile Gly Asp Gln Tyr Ala Asp Leu Phe
355 360 365
Leu Ala Ala Lys Asn Leu Ser Asp Ala Ile Leu Leu Ser Asp Ile Leu
370 375 380
Arg Val Asn Thr Glu Ile Thr Lys Ala Pro Leu Ser Ala Ser Met Ile
385 390 395 400
Lys Arg Tyr Asp Glu His His Gln Asp Leu Thr Leu Leu Lys Ala Leu
405 410 415
Val Arg Gln Gln Leu Pro Glu Lys Tyr Lys Glu Ile Phe Phe Asp Gln
420 425 430
Ser Lys Asn Gly Tyr Ala Gly Tyr Ile Asp Gly Gly Ala Ser Gln Glu
435 440 445
Glu Phe Tyr Lys Phe Ile Lys Pro Ile Leu Glu Lys Met Asp Gly Thr
450 455 460
Glu Glu Leu Leu Val Lys Leu Asn Arg Glu Asp Leu Leu Arg Lys Gln
465 470 475 480
Arg Thr Phe Asp Asn Gly Ser Ile Pro His Gln Ile His Leu Gly Glu
485 490 495
Leu His Ala Ile Leu Arg Arg Gln Glu Asp Phe Tyr Pro Phe Leu Lys
500 505 510
Asp Asn Arg Glu Lys Ile Glu Lys Ile Leu Thr Phe Arg Ile Pro Tyr
515 520 525
Tyr Val Gly Pro Leu Ala Arg Gly Asn Ser Arg Phe Ala Trp Met Thr
530 535 540
Arg Lys Ser Glu Glu Thr Ile Thr Pro Trp Asn Phe Glu Glu Val Val
545 550 555 560
Asp Lys Gly Ala Ser Ala Gln Ser Phe Ile Glu Arg Met Thr Asn Phe
565 570 575
Asp Lys Asn Leu Pro Asn Glu Lys Val Leu Pro Lys His Ser Leu Leu
580 585 590
Tyr Glu Tyr Phe Thr Val Tyr Asn Glu Leu Thr Lys Val Lys Tyr Val
595 600 605
Thr Glu Gly Met Arg Lys Pro Ala Phe Leu Ser Gly Glu Gln Lys Lys
610 615 620
Ala Ile Val Asp Leu Leu Phe Lys Thr Asn Arg Lys Val Thr Val Lys
625 630 635 640
Gln Leu Lys Glu Asp Tyr Phe Lys Lys Ile Glu Cys Phe Asp Ser Val
645 650 655
Glu Ile Ser Gly Val Glu Asp Arg Phe Asn Ala Ser Leu Gly Thr Tyr
660 665 670
His Asp Leu Leu Lys Ile Ile Lys Asp Lys Asp Phe Leu Asp Asn Glu
675 680 685
Glu Asn Glu Asp Ile Leu Glu Asp Ile Val Leu Thr Leu Thr Leu Phe
690 695 700
Glu Asp Arg Glu Met Ile Glu Glu Arg Leu Lys Thr Tyr Ala His Leu
705 710 715 720
Phe Asp Asp Lys Val Met Lys Gln Leu Lys Arg Arg Arg Tyr Thr Gly
725 730 735
Trp Gly Arg Leu Ser Arg Lys Leu Ile Asn Gly Ile Arg Asp Lys Gln
740 745 750
Ser Gly Lys Thr Ile Leu Asp Phe Leu Lys Ser Asp Gly Phe Ala Asn
755 760 765
Arg Asn Phe Met Gln Leu Ile His Asp Asp Ser Leu Thr Phe Lys Glu
770 775 780
Asp Ile Gln Lys Ala Gln Val Ser Gly Gln Gly Asp Ser Leu His Glu
785 790 795 800
His Ile Ala Asn Leu Ala Gly Ser Pro Ala Ile Lys Lys Gly Ile Leu
805 810 815
Gln Thr Val Lys Val Val Asp Glu Leu Val Lys Val Met Gly Arg His
820 825 830
Lys Pro Glu Asn Ile Val Ile Glu Met Ala Arg Glu Asn Gln Thr Thr
835 840 845
Gln Lys Gly Gln Lys Asn Ser Arg Glu Arg Met Lys Arg Ile Glu Glu
850 855 860
Gly Ile Lys Glu Leu Gly Ser Gln Ile Leu Lys Glu His Pro Val Glu
865 870 875 880
Asn Thr Gln Leu Gln Asn Glu Lys Leu Tyr Leu Tyr Tyr Leu Gln Asn
885 890 895
Gly Arg Asp Met Tyr Val Asp Gln Glu Leu Asp Ile Asn Arg Leu Ser
900 905 910
Asp Tyr Asp Val Asp Ala Ile Val Pro Gln Ser Phe Leu Lys Asp Asp
915 920 925
Ser Ile Asp Asn Lys Val Leu Thr Arg Ser Asp Lys Asn Arg Gly Lys
930 935 940
Ser Asp Asn Val Pro Ser Glu Glu Val Val Lys Lys Met Lys Asn Tyr
945 950 955 960
Trp Arg Gln Leu Leu Asn Ala Lys Leu Ile Thr Gln Arg Lys Phe Asp
965 970 975
Asn Leu Thr Lys Ala Glu Arg Gly Gly Leu Ser Glu Leu Asp Lys Ala
980 985 990
Gly Phe Ile Lys Arg Gln Leu Val Glu Thr Arg Gln Ile Thr Lys His
995 1000 1005
Val Ala Gln Ile Leu Asp Ser Arg Met Asn Thr Lys Tyr Asp Glu
1010 1015 1020
Asn Asp Lys Leu Ile Arg Glu Val Lys Val Ile Thr Leu Lys Ser
1025 1030 1035
Lys Leu Val Ser Asp Phe Arg Lys Asp Phe Gln Phe Tyr Lys Val
1040 1045 1050
Arg Glu Ile Asn Asn Tyr His His Ala His Asp Ala Tyr Leu Asn
1055 1060 1065
Ala Val Val Gly Thr Ala Leu Ile Lys Lys Tyr Pro Lys Leu Glu
1070 1075 1080
Ser Glu Phe Val Tyr Gly Asp Tyr Lys Val Tyr Asp Val Arg Lys
1085 1090 1095
Met Ile Ala Lys Ser Glu Gln Glu Ile Gly Lys Ala Thr Ala Lys
1100 1105 1110
Tyr Phe Phe Tyr Ser Asn Ile Met Asn Phe Phe Lys Thr Glu Ile
1115 1120 1125
Thr Leu Ala Asn Gly Glu Ile Arg Lys Arg Pro Leu Ile Glu Thr
1130 1135 1140
Asn Gly Glu Thr Gly Glu Ile Val Trp Asp Lys Gly Arg Asp Phe
1145 1150 1155
Ala Thr Val Arg Lys Val Leu Ser Met Pro Gln Val Asn Ile Val
1160 1165 1170
Lys Lys Thr Glu Val Gln Thr Gly Gly Phe Ser Lys Glu Ser Ile
1175 1180 1185
Leu Pro Lys Arg Asn Ser Asp Lys Leu Ile Ala Arg Lys Lys Asp
1190 1195 1200
Trp Asp Pro Lys Lys Tyr Gly Gly Phe Asp Ser Pro Thr Val Ala
1205 1210 1215
Tyr Ser Val Leu Val Val Ala Lys Val Glu Lys Gly Lys Ser Lys
1220 1225 1230
Lys Leu Lys Ser Val Lys Glu Leu Leu Gly Ile Thr Ile Met Glu
1235 1240 1245
Arg Ser Ser Phe Glu Lys Asn Pro Ile Asp Phe Leu Glu Ala Lys
1250 1255 1260
Gly Tyr Lys Glu Val Lys Lys Asp Leu Ile Ile Lys Leu Pro Lys
1265 1270 1275
Tyr Ser Leu Phe Glu Leu Glu Asn Gly Arg Lys Arg Met Leu Ala
1280 1285 1290
Ser Ala Gly Glu Leu Gln Lys Gly Asn Glu Leu Ala Leu Pro Ser
1295 1300 1305
Lys Tyr Val Asn Phe Leu Tyr Leu Ala Ser His Tyr Glu Lys Leu
1310 1315 1320
Lys Gly Ser Pro Glu Asp Asn Glu Gln Lys Gln Leu Phe Val Glu
1325 1330 1335
Gln His Lys His Tyr Leu Asp Glu Ile Ile Glu Gln Ile Ser Glu
1340 1345 1350
Phe Ser Lys Arg Val Ile Leu Ala Asp Ala Asn Leu Asp Lys Val
1355 1360 1365
Leu Ser Ala Tyr Asn Lys His Arg Asp Lys Pro Ile Arg Glu Gln
1370 1375 1380
Ala Glu Asn Ile Ile His Leu Phe Thr Leu Thr Asn Leu Gly Ala
1385 1390 1395
Pro Ala Ala Phe Lys Tyr Phe Asp Thr Thr Ile Asp Arg Lys Arg
1400 1405 1410
Tyr Thr Ser Thr Lys Glu Val Leu Asp Ala Thr Leu Ile His Gln
1415 1420 1425
Ser Ile Thr Gly Leu Tyr Glu Thr Arg Ile Asp Leu Ser Gln Leu
1430 1435 1440
Gly Gly Asp Ser Arg Ala Asp Tyr Pro Tyr Asp Val Pro Asp Tyr
1445 1450 1455
Ala Ser Gly Ser Pro Lys Lys Lys Arg Lys Val Ser Pro Gly Ser
1460 1465 1470
Gly Ser Glu Thr Pro Gly Thr Ser Glu Ser Ala Thr Pro Glu Ser
1475 1480 1485
Asn His Asp Gln Glu Phe Asp Pro Pro Lys Val Tyr Pro Pro Val
1490 1495 1500
Pro Ala Glu Lys Arg Lys Pro Ile Arg Val Leu Ser Leu Phe Asp
1505 1510 1515
Gly Ile Ala Thr Gly Leu Leu Val Leu Lys Asp Leu Gly Ile Gln
1520 1525 1530
Val Asp Arg Tyr Ile Ala Ser Glu Val Cys Glu Asp Ser Ile Thr
1535 1540 1545
Val Gly Met Val Arg His Gln Gly Lys Ile Met Tyr Val Gly Asp
1550 1555 1560
Val Arg Ser Val Thr Gln Lys His Ile Gln Glu Trp Gly Pro Phe
1565 1570 1575
Asp Leu Val Ile Gly Gly Ser Pro Cys Asn Asp Leu Ser Ile Val
1580 1585 1590
Asn Pro Ala Arg Lys Gly Leu Tyr Glu Gly Thr Gly Arg Leu Phe
1595 1600 1605
Phe Glu Phe Tyr Arg Leu Leu His Asp Ala Arg Pro Lys Glu Gly
1610 1615 1620
Asp Asp Arg Pro Phe Phe Trp Leu Phe Glu Asn Val Val Ala Met
1625 1630 1635
Gly Val Ser Asp Lys Arg Asp Ile Ser Arg Phe Leu Glu Ser Asn
1640 1645 1650
Pro Val Met Ile Asp Ala Lys Glu Val Ser Ala Ala His Arg Ala
1655 1660 1665
Arg Tyr Phe Trp Gly Asn Leu Pro Gly Met Asn Arg Pro Leu Ala
1670 1675 1680
Ser Thr Val Asn Asp Lys Leu Glu Leu Gln Glu Cys Leu Glu His
1685 1690 1695
Gly Arg Ile Ala Lys Phe Ser Lys Val Arg Thr Ile Thr Thr Arg
1700 1705 1710
Ser Asn Ser Ile Lys Gln Gly Lys Asp Gln His Phe Pro Val Phe
1715 1720 1725
Met Asn Glu Lys Glu Asp Ile Leu Trp Cys Thr Glu Met Glu Arg
1730 1735 1740
Val Phe Gly Phe Pro Val His Tyr Thr Asp Val Ser Asn Met Ser
1745 1750 1755
Arg Leu Ala Arg Gln Arg Leu Leu Gly Arg Ser Trp Ser Val Pro
1760 1765 1770
Val Ile Arg His Leu Phe Ala Pro Leu Lys Glu Tyr Phe Ala Cys
1775 1780 1785
Val Ser Ser Gly Asn Ser Asn Ala Asn Ser Arg Gly Pro Ser Phe
1790 1795 1800
Ser Ser Gly Leu Val Pro Leu Ser Leu Arg Gly Ser His Met Gly
1805 1810 1815
Pro Met Glu Ile Tyr Lys Thr Val Ser Ala Trp Lys Arg Gln Pro
1820 1825 1830
Val Arg Val Leu Ser Leu Phe Arg Asn Ile Asp Lys Val Leu Lys
1835 1840 1845
Ser Leu Gly Phe Leu Glu Ser Gly Ser Gly Ser Gly Gly Gly Thr
1850 1855 1860
Leu Lys Tyr Val Glu Asp Val Thr Asn Val Val Arg Arg Asp Val
1865 1870 1875
Glu Lys Trp Gly Pro Phe Asp Leu Val Tyr Gly Ser Thr Gln Pro
1880 1885 1890
Leu Gly Ser Ser Cys Asp Arg Cys Pro Gly Trp Tyr Met Phe Gln
1895 1900 1905
Phe His Arg Ile Leu Gln Tyr Ala Leu Pro Arg Gln Glu Ser Gln
1910 1915 1920
Arg Pro Phe Phe Trp Ile Phe Met Asp Asn Leu Leu Leu Thr Glu
1925 1930 1935
Asp Asp Gln Glu Thr Thr Thr Arg Phe Leu Gln Thr Glu Ala Val
1940 1945 1950
Thr Leu Gln Asp Val Arg Gly Arg Asp Tyr Gln Asn Ala Met Arg
1955 1960 1965
Val Trp Ser Asn Ile Pro Gly Leu Lys Ser Lys His Ala Pro Leu
1970 1975 1980
Thr Pro Lys Glu Glu Glu Tyr Leu Gln Ala Gln Val Arg Ser Arg
1985 1990 1995
Ser Lys Leu Asp Ala Pro Lys Val Asp Leu Leu Val Lys Asn Cys
2000 2005 2010
Leu Leu Pro Leu Arg Glu Tyr Phe Lys Tyr Phe Ser Gln Asn Ser
2015 2020 2025
Leu Pro Leu Ser Arg Ala Asp Pro Lys Lys Lys Arg Lys Val Gly
2030 2035 2040
Ser Gly Ala Thr Asn Phe Ser Leu Leu Lys Gln Ala Gly Asp Val
2045 2050 2055
Glu Glu Asn Pro Gly Pro Gly Ser Gly Ala Thr Asn Phe Ser Leu
2060 2065 2070
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Ser Glu
2075 2080 2085
Leu Ile Lys Glu Asn Met His Met Lys Leu Tyr Met Glu Gly Thr
2090 2095 2100
Val Asp Asn His His Phe Lys Cys Thr Ser Glu Gly Glu Gly Lys
2105 2110 2115
Pro Tyr Glu Gly Thr Gln Thr Met Arg Ile Lys Val Val Glu Gly
2120 2125 2130
Gly Pro Leu Pro Phe Ala Phe Asp Ile Leu Ala Thr Ser Phe Leu
2135 2140 2145
Tyr Gly Ser Lys Thr Phe Ile Asn His Thr Gln Gly Ile Pro Asp
2150 2155 2160
Phe Phe Lys Gln Ser Phe Pro Glu Gly Phe Thr Trp Glu Arg Val
2165 2170 2175
Thr Thr Tyr Glu Asp Gly Gly Val Leu Thr Ala Thr Gln Asp Thr
2180 2185 2190
Ser Leu Gln Asp Gly Cys Leu Ile Tyr Asn Val Lys Ile Arg Gly
2195 2200 2205
Val Asn Phe Thr Ser Asn Gly Pro Val Met Gln Lys Lys Thr Leu
2210 2215 2220
Gly Trp Glu Ala Phe Thr Glu Thr Leu Tyr Pro Ala Asp Gly Gly
2225 2230 2235
Leu Glu Gly Arg Asn Asp Met Ala Leu Lys Leu Val Gly Gly Ser
2240 2245 2250
His Leu Ile Ala Asn Ile Lys Thr Thr Tyr Arg Ser Lys Lys Pro
2255 2260 2265
Ala Lys Asn Leu Lys Met Pro Gly Val Tyr Tyr Val Asp Tyr Arg
2270 2275 2280
Leu Glu Arg Ile Lys Glu Ala Asn Asn Glu Thr Tyr Val Glu Gln
2285 2290 2295
His Glu Val Ala Val Ala Arg Tyr Cys Asp Leu Pro Ser Lys Leu
2300 2305 2310
Gly His Lys Leu Asn
2315
<210> 5
<211> 2360
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетический полипептид
<400> 5
Asp Ala Lys Ser Leu Thr Ala Trp Ser Arg Thr Leu Val Thr Phe Lys
1. 5 10 15
Asp Val Phe Val Asp Phe Thr Arg Glu Glu Trp Lys Leu Leu Asp Thr
20 25 30
Ala Gln Gln Ile Val Tyr Arg Asn Val Met Leu Glu Asn Tyr Lys Asn
35 40 45
Leu Val Ser Leu Gly Tyr Gln Leu Thr Lys Pro Asp Val Ile Leu Arg
50 55 60
Leu Glu Lys Gly Glu Glu Pro Gly Gly Ser Gly Gly Gly Ser Met Asp
65 70 75 80
Lys Lys Tyr Ser Ile Gly Leu Ala Ile Gly Thr Asn Ser Val Gly Trp
85 90 95
Ala Val Ile Thr Asp Glu Tyr Lys Val Pro Ser Lys Lys Phe Lys Val
100 105 110
Leu Gly Asn Thr Asp Arg His Ser Ile Lys Lys Asn Leu Ile Gly Ala
115 120 125
Leu Leu Phe Asp Ser Gly Glu Thr Ala Glu Ala Thr Arg Leu Lys Arg
130 135 140
Thr Ala Arg Arg Arg Tyr Thr Arg Arg Lys Asn Arg Ile Cys Tyr Leu
145 150 155 160
Gln Glu Ile Phe Ser Asn Glu Met Ala Lys Val Asp Asp Ser Phe Phe
165 170 175
His Arg Leu Glu Glu Ser Phe Leu Val Glu Glu Asp Lys Lys His Glu
180 185 190
Arg His Pro Ile Phe Gly Asn Ile Val Asp Glu Val Ala Tyr His Glu
195 200 205
Lys Tyr Pro Thr Ile Tyr His Leu Arg Lys Lys Leu Val Asp Ser Thr
210 215 220
Asp Lys Ala Asp Leu Arg Leu Ile Tyr Leu Ala Leu Ala His Met Ile
225 230 235 240
Lys Phe Arg Gly His Phe Leu Ile Glu Gly Asp Leu Asn Pro Asp Asn
245 250 255
Ser Asp Val Asp Lys Leu Phe Ile Gln Leu Val Gln Thr Tyr Asn Gln
260 265 270
Leu Phe Glu Glu Asn Pro Ile Asn Ala Ser Gly Val Asp Ala Lys Ala
275 280 285
Ile Leu Ser Ala Arg Leu Ser Lys Ser Arg Arg Leu Glu Asn Leu Ile
290 295 300
Ala Gln Leu Pro Gly Glu Lys Lys Asn Gly Leu Phe Gly Asn Leu Ile
305 310 315 320
Ala Leu Ser Leu Gly Leu Thr Pro Asn Phe Lys Ser Asn Phe Asp Leu
325 330 335
Ala Glu Asp Ala Lys Leu Gln Leu Ser Lys Asp Thr Tyr Asp Asp Asp
340 345 350
Leu Asp Asn Leu Leu Ala Gln Ile Gly Asp Gln Tyr Ala Asp Leu Phe
355 360 365
Leu Ala Ala Lys Asn Leu Ser Asp Ala Ile Leu Leu Ser Asp Ile Leu
370 375 380
Arg Val Asn Thr Glu Ile Thr Lys Ala Pro Leu Ser Ala Ser Met Ile
385 390 395 400
Lys Arg Tyr Asp Glu His His Gln Asp Leu Thr Leu Leu Lys Ala Leu
405 410 415
Val Arg Gln Gln Leu Pro Glu Lys Tyr Lys Glu Ile Phe Phe Asp Gln
420 425 430
Ser Lys Asn Gly Tyr Ala Gly Tyr Ile Asp Gly Gly Ala Ser Gln Glu
435 440 445
Glu Phe Tyr Lys Phe Ile Lys Pro Ile Leu Glu Lys Met Asp Gly Thr
450 455 460
Glu Glu Leu Leu Val Lys Leu Asn Arg Glu Asp Leu Leu Arg Lys Gln
465 470 475 480
Arg Thr Phe Asp Asn Gly Ser Ile Pro His Gln Ile His Leu Gly Glu
485 490 495
Leu His Ala Ile Leu Arg Arg Gln Glu Asp Phe Tyr Pro Phe Leu Lys
500 505 510
Asp Asn Arg Glu Lys Ile Glu Lys Ile Leu Thr Phe Arg Ile Pro Tyr
515 520 525
Tyr Val Gly Pro Leu Ala Arg Gly Asn Ser Arg Phe Ala Trp Met Thr
530 535 540
Arg Lys Ser Glu Glu Thr Ile Thr Pro Trp Asn Phe Glu Glu Val Val
545 550 555 560
Asp Lys Gly Ala Ser Ala Gln Ser Phe Ile Glu Arg Met Thr Asn Phe
565 570 575
Asp Lys Asn Leu Pro Asn Glu Lys Val Leu Pro Lys His Ser Leu Leu
580 585 590
Tyr Glu Tyr Phe Thr Val Tyr Asn Glu Leu Thr Lys Val Lys Tyr Val
595 600 605
Thr Glu Gly Met Arg Lys Pro Ala Phe Leu Ser Gly Glu Gln Lys Lys
610 615 620
Ala Ile Val Asp Leu Leu Phe Lys Thr Asn Arg Lys Val Thr Val Lys
625 630 635 640
Gln Leu Lys Glu Asp Tyr Phe Lys Lys Ile Glu Cys Phe Asp Ser Val
645 650 655
Glu Ile Ser Gly Val Glu Asp Arg Phe Asn Ala Ser Leu Gly Thr Tyr
660 665 670
His Asp Leu Leu Lys Ile Ile Lys Asp Lys Asp Phe Leu Asp Asn Glu
675 680 685
Glu Asn Glu Asp Ile Leu Glu Asp Ile Val Leu Thr Leu Thr Leu Phe
690 695 700
Glu Asp Arg Glu Met Ile Glu Glu Arg Leu Lys Thr Tyr Ala His Leu
705 710 715 720
Phe Asp Asp Lys Val Met Lys Gln Leu Lys Arg Arg Arg Tyr Thr Gly
725 730 735
Trp Gly Arg Leu Ser Arg Lys Leu Ile Asn Gly Ile Arg Asp Lys Gln
740 745 750
Ser Gly Lys Thr Ile Leu Asp Phe Leu Lys Ser Asp Gly Phe Ala Asn
755 760 765
Arg Asn Phe Met Gln Leu Ile His Asp Asp Ser Leu Thr Phe Lys Glu
770 775 780
Asp Ile Gln Lys Ala Gln Val Ser Gly Gln Gly Asp Ser Leu His Glu
785 790 795 800
His Ile Ala Asn Leu Ala Gly Ser Pro Ala Ile Lys Lys Gly Ile Leu
805 810 815
Gln Thr Val Lys Val Val Asp Glu Leu Val Lys Val Met Gly Arg His
820 825 830
Lys Pro Glu Asn Ile Val Ile Glu Met Ala Arg Glu Asn Gln Thr Thr
835 840 845
Gln Lys Gly Gln Lys Asn Ser Arg Glu Arg Met Lys Arg Ile Glu Glu
850 855 860
Gly Ile Lys Glu Leu Gly Ser Gln Ile Leu Lys Glu His Pro Val Glu
865 870 875 880
Asn Thr Gln Leu Gln Asn Glu Lys Leu Tyr Leu Tyr Tyr Leu Gln Asn
885 890 895
Gly Arg Asp Met Tyr Val Asp Gln Glu Leu Asp Ile Asn Arg Leu Ser
900 905 910
Asp Tyr Asp Val Asp Ala Ile Val Pro Gln Ser Phe Leu Lys Asp Asp
915 920 925
Ser Ile Asp Asn Lys Val Leu Thr Arg Ser Asp Lys Asn Arg Gly Lys
930 935 940
Ser Asp Asn Val Pro Ser Glu Glu Val Val Lys Lys Met Lys Asn Tyr
945 950 955 960
Trp Arg Gln Leu Leu Asn Ala Lys Leu Ile Thr Gln Arg Lys Phe Asp
965 970 975
Asn Leu Thr Lys Ala Glu Arg Gly Gly Leu Ser Glu Leu Asp Lys Ala
980 985 990
Gly Phe Ile Lys Arg Gln Leu Val Glu Thr Arg Gln Ile Thr Lys His
995 1000 1005
Val Ala Gln Ile Leu Asp Ser Arg Met Asn Thr Lys Tyr Asp Glu
1010 1015 1020
Asn Asp Lys Leu Ile Arg Glu Val Lys Val Ile Thr Leu Lys Ser
1025 1030 1035
Lys Leu Val Ser Asp Phe Arg Lys Asp Phe Gln Phe Tyr Lys Val
1040 1045 1050
Arg Glu Ile Asn Asn Tyr His His Ala His Asp Ala Tyr Leu Asn
1055 1060 1065
Ala Val Val Gly Thr Ala Leu Ile Lys Lys Tyr Pro Lys Leu Glu
1070 1075 1080
Ser Glu Phe Val Tyr Gly Asp Tyr Lys Val Tyr Asp Val Arg Lys
1085 1090 1095
Met Ile Ala Lys Ser Glu Gln Glu Ile Gly Lys Ala Thr Ala Lys
1100 1105 1110
Tyr Phe Phe Tyr Ser Asn Ile Met Asn Phe Phe Lys Thr Glu Ile
1115 1120 1125
Thr Leu Ala Asn Gly Glu Ile Arg Lys Arg Pro Leu Ile Glu Thr
1130 1135 1140
Asn Gly Glu Thr Gly Glu Ile Val Trp Asp Lys Gly Arg Asp Phe
1145 1150 1155
Ala Thr Val Arg Lys Val Leu Ser Met Pro Gln Val Asn Ile Val
1160 1165 1170
Lys Lys Thr Glu Val Gln Thr Gly Gly Phe Ser Lys Glu Ser Ile
1175 1180 1185
Leu Pro Lys Arg Asn Ser Asp Lys Leu Ile Ala Arg Lys Lys Asp
1190 1195 1200
Trp Asp Pro Lys Lys Tyr Gly Gly Phe Asp Ser Pro Thr Val Ala
1205 1210 1215
Tyr Ser Val Leu Val Val Ala Lys Val Glu Lys Gly Lys Ser Lys
1220 1225 1230
Lys Leu Lys Ser Val Lys Glu Leu Leu Gly Ile Thr Ile Met Glu
1235 1240 1245
Arg Ser Ser Phe Glu Lys Asn Pro Ile Asp Phe Leu Glu Ala Lys
1250 1255 1260
Gly Tyr Lys Glu Val Lys Lys Asp Leu Ile Ile Lys Leu Pro Lys
1265 1270 1275
Tyr Ser Leu Phe Glu Leu Glu Asn Gly Arg Lys Arg Met Leu Ala
1280 1285 1290
Ser Ala Gly Glu Leu Gln Lys Gly Asn Glu Leu Ala Leu Pro Ser
1295 1300 1305
Lys Tyr Val Asn Phe Leu Tyr Leu Ala Ser His Tyr Glu Lys Leu
1310 1315 1320
Lys Gly Ser Pro Glu Asp Asn Glu Gln Lys Gln Leu Phe Val Glu
1325 1330 1335
Gln His Lys His Tyr Leu Asp Glu Ile Ile Glu Gln Ile Ser Glu
1340 1345 1350
Phe Ser Lys Arg Val Ile Leu Ala Asp Ala Asn Leu Asp Lys Val
1355 1360 1365
Leu Ser Ala Tyr Asn Lys His Arg Asp Lys Pro Ile Arg Glu Gln
1370 1375 1380
Ala Glu Asn Ile Ile His Leu Phe Thr Leu Thr Asn Leu Gly Ala
1385 1390 1395
Pro Ala Ala Phe Lys Tyr Phe Asp Thr Thr Ile Asp Arg Lys Arg
1400 1405 1410
Tyr Thr Ser Thr Lys Glu Val Leu Asp Ala Thr Leu Ile His Gln
1415 1420 1425
Ser Ile Thr Gly Leu Tyr Glu Thr Arg Ile Asp Leu Ser Gln Leu
1430 1435 1440
Gly Gly Asp Ser Arg Ala Asp Tyr Pro Tyr Asp Val Pro Asp Tyr
1445 1450 1455
Ala Ser Gly Ser Pro Lys Lys Lys Arg Lys Val Ser Pro Gly Gly
1460 1465 1470
Gly Pro Ser Ser Gly Ala Pro Pro Pro Ser Gly Gly Ser Pro Ala
1475 1480 1485
Gly Ser Pro Thr Ser Thr Glu Glu Gly Thr Ser Glu Ser Ala Thr
1490 1495 1500
Pro Glu Ser Gly Pro Gly Thr Ser Thr Glu Pro Ser Glu Gly Ser
1505 1510 1515
Ala Pro Gly Ser Pro Ala Gly Ser Pro Thr Ser Thr Glu Glu Gly
1520 1525 1530
Thr Ser Thr Glu Pro Ser Glu Gly Ser Ala Pro Gly Thr Ser Thr
1535 1540 1545
Glu Pro Ser Glu Asn His Asp Gln Glu Phe Asp Pro Pro Lys Val
1550 1555 1560
Tyr Pro Pro Val Pro Ala Glu Lys Arg Lys Pro Ile Arg Val Leu
1565 1570 1575
Ser Leu Phe Asp Gly Ile Ala Thr Gly Leu Leu Val Leu Lys Asp
1580 1585 1590
Leu Gly Ile Gln Val Asp Arg Tyr Ile Ala Ser Glu Val Cys Glu
1595 1600 1605
Asp Ser Ile Thr Val Gly Met Val Arg His Gln Gly Lys Ile Met
1610 1615 1620
Tyr Val Gly Asp Val Arg Ser Val Thr Gln Lys His Ile Gln Glu
1625 1630 1635
Trp Gly Pro Phe Asp Leu Val Ile Gly Gly Ser Pro Cys Asn Asp
1640 1645 1650
Leu Ser Ile Val Asn Pro Ala Arg Lys Gly Leu Tyr Glu Gly Thr
1655 1660 1665
Gly Arg Leu Phe Phe Glu Phe Tyr Arg Leu Leu His Asp Ala Arg
1670 1675 1680
Pro Lys Glu Gly Asp Asp Arg Pro Phe Phe Trp Leu Phe Glu Asn
1685 1690 1695
Val Val Ala Met Gly Val Ser Asp Lys Arg Asp Ile Ser Arg Phe
1700 1705 1710
Leu Glu Ser Asn Pro Val Met Ile Asp Ala Lys Glu Val Ser Ala
1715 1720 1725
Ala His Arg Ala Arg Tyr Phe Trp Gly Asn Leu Pro Gly Met Asn
1730 1735 1740
Arg Pro Leu Ala Ser Thr Val Asn Asp Lys Leu Glu Leu Gln Glu
1745 1750 1755
Cys Leu Glu His Gly Arg Ile Ala Lys Phe Ser Lys Val Arg Thr
1760 1765 1770
Ile Thr Thr Arg Ser Asn Ser Ile Lys Gln Gly Lys Asp Gln His
1775 1780 1785
Phe Pro Val Phe Met Asn Glu Lys Glu Asp Ile Leu Trp Cys Thr
1790 1795 1800
Glu Met Glu Arg Val Phe Gly Phe Pro Val His Tyr Thr Asp Val
1805 1810 1815
Ser Asn Met Ser Arg Leu Ala Arg Gln Arg Leu Leu Gly Arg Ser
1820 1825 1830
Trp Ser Val Pro Val Ile Arg His Leu Phe Ala Pro Leu Lys Glu
1835 1840 1845
Tyr Phe Ala Cys Val Ser Ser Gly Asn Ser Asn Ala Asn Ser Arg
1850 1855 1860
Gly Pro Ser Phe Ser Ser Gly Leu Val Pro Leu Ser Leu Arg Gly
1865 1870 1875
Ser His Met Gly Pro Met Glu Ile Tyr Lys Thr Val Ser Ala Trp
1880 1885 1890
Lys Arg Gln Pro Val Arg Val Leu Ser Leu Phe Arg Asn Ile Asp
1895 1900 1905
Lys Val Leu Lys Ser Leu Gly Phe Leu Glu Ser Gly Ser Gly Ser
1910 1915 1920
Gly Gly Gly Thr Leu Lys Tyr Val Glu Asp Val Thr Asn Val Val
1925 1930 1935
Arg Arg Asp Val Glu Lys Trp Gly Pro Phe Asp Leu Val Tyr Gly
1940 1945 1950
Ser Thr Gln Pro Leu Gly Ser Ser Cys Asp Arg Cys Pro Gly Trp
1955 1960 1965
Tyr Met Phe Gln Phe His Arg Ile Leu Gln Tyr Ala Leu Pro Arg
1970 1975 1980
Gln Glu Ser Gln Arg Pro Phe Phe Trp Ile Phe Met Asp Asn Leu
1985 1990 1995
Leu Leu Thr Glu Asp Asp Gln Glu Thr Thr Thr Arg Phe Leu Gln
2000 2005 2010
Thr Glu Ala Val Thr Leu Gln Asp Val Arg Gly Arg Asp Tyr Gln
2015 2020 2025
Asn Ala Met Arg Val Trp Ser Asn Ile Pro Gly Leu Lys Ser Lys
2030 2035 2040
His Ala Pro Leu Thr Pro Lys Glu Glu Glu Tyr Leu Gln Ala Gln
2045 2050 2055
Val Arg Ser Arg Ser Lys Leu Asp Ala Pro Lys Val Asp Leu Leu
2060 2065 2070
Val Lys Asn Cys Leu Leu Pro Leu Arg Glu Tyr Phe Lys Tyr Phe
2075 2080 2085
Ser Gln Asn Ser Leu Pro Leu Ser Arg Ala Asp Pro Lys Lys Lys
2090 2095 2100
Arg Lys Val Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu Lys Gln
2105 2110 2115
Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Ser Glu Leu Ile Lys
2120 2125 2130
Glu Asn Met His Met Lys Leu Tyr Met Glu Gly Thr Val Asp Asn
2135 2140 2145
His His Phe Lys Cys Thr Ser Glu Gly Glu Gly Lys Pro Tyr Glu
2150 2155 2160
Gly Thr Gln Thr Met Arg Ile Lys Val Val Glu Gly Gly Pro Leu
2165 2170 2175
Pro Phe Ala Phe Asp Ile Leu Ala Thr Ser Phe Leu Tyr Gly Ser
2180 2185 2190
Lys Thr Phe Ile Asn His Thr Gln Gly Ile Pro Asp Phe Phe Lys
2195 2200 2205
Gln Ser Phe Pro Glu Gly Phe Thr Trp Glu Arg Val Thr Thr Tyr
2210 2215 2220
Glu Asp Gly Gly Val Leu Thr Ala Thr Gln Asp Thr Ser Leu Gln
2225 2230 2235
Asp Gly Cys Leu Ile Tyr Asn Val Lys Ile Arg Gly Val Asn Phe
2240 2245 2250
Thr Ser Asn Gly Pro Val Met Gln Lys Lys Thr Leu Gly Trp Glu
2255 2260 2265
Ala Phe Thr Glu Thr Leu Tyr Pro Ala Asp Gly Gly Leu Glu Gly
2270 2275 2280
Arg Asn Asp Met Ala Leu Lys Leu Val Gly Gly Ser His Leu Ile
2285 2290 2295
Ala Asn Ile Lys Thr Thr Tyr Arg Ser Lys Lys Pro Ala Lys Asn
2300 2305 2310
Leu Lys Met Pro Gly Val Tyr Tyr Val Asp Tyr Arg Leu Glu Arg
2315 2320 2325
Ile Lys Glu Ala Asn Asn Glu Thr Tyr Val Glu Gln His Glu Val
2330 2335 2340
Ala Val Ala Arg Tyr Cys Asp Leu Pro Ser Lys Leu Gly His Lys
2345 2350 2355
Leu Asn
2360
<210> 6
<211> 2382
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетический полипептид
<400> 6
Asp Ala Lys Ser Leu Thr Ala Trp Ser Arg Thr Leu Val Thr Phe Lys
1. 5 10 15
Asp Val Phe Val Asp Phe Thr Arg Glu Glu Trp Lys Leu Leu Asp Thr
20 25 30
Ala Gln Gln Ile Val Tyr Arg Asn Val Met Leu Glu Asn Tyr Lys Asn
35 40 45
Leu Val Ser Leu Gly Tyr Gln Leu Thr Lys Pro Asp Val Ile Leu Arg
50 55 60
Leu Glu Lys Gly Glu Glu Pro Gly Gly Ser Gly Gly Gly Ser Met Asp
65 70 75 80
Lys Lys Tyr Ser Ile Gly Leu Ala Ile Gly Thr Asn Ser Val Gly Trp
85 90 95
Ala Val Ile Thr Asp Glu Tyr Lys Val Pro Ser Lys Lys Phe Lys Val
100 105 110
Leu Gly Asn Thr Asp Arg His Ser Ile Lys Lys Asn Leu Ile Gly Ala
115 120 125
Leu Leu Phe Asp Ser Gly Glu Thr Ala Glu Ala Thr Arg Leu Lys Arg
130 135 140
Thr Ala Arg Arg Arg Tyr Thr Arg Arg Lys Asn Arg Ile Cys Tyr Leu
145 150 155 160
Gln Glu Ile Phe Ser Asn Glu Met Ala Lys Val Asp Asp Ser Phe Phe
165 170 175
His Arg Leu Glu Glu Ser Phe Leu Val Glu Glu Asp Lys Lys His Glu
180 185 190
Arg His Pro Ile Phe Gly Asn Ile Val Asp Glu Val Ala Tyr His Glu
195 200 205
Lys Tyr Pro Thr Ile Tyr His Leu Arg Lys Lys Leu Val Asp Ser Thr
210 215 220
Asp Lys Ala Asp Leu Arg Leu Ile Tyr Leu Ala Leu Ala His Met Ile
225 230 235 240
Lys Phe Arg Gly His Phe Leu Ile Glu Gly Asp Leu Asn Pro Asp Asn
245 250 255
Ser Asp Val Asp Lys Leu Phe Ile Gln Leu Val Gln Thr Tyr Asn Gln
260 265 270
Leu Phe Glu Glu Asn Pro Ile Asn Ala Ser Gly Val Asp Ala Lys Ala
275 280 285
Ile Leu Ser Ala Arg Leu Ser Lys Ser Arg Arg Leu Glu Asn Leu Ile
290 295 300
Ala Gln Leu Pro Gly Glu Lys Lys Asn Gly Leu Phe Gly Asn Leu Ile
305 310 315 320
Ala Leu Ser Leu Gly Leu Thr Pro Asn Phe Lys Ser Asn Phe Asp Leu
325 330 335
Ala Glu Asp Ala Lys Leu Gln Leu Ser Lys Asp Thr Tyr Asp Asp Asp
340 345 350
Leu Asp Asn Leu Leu Ala Gln Ile Gly Asp Gln Tyr Ala Asp Leu Phe
355 360 365
Leu Ala Ala Lys Asn Leu Ser Asp Ala Ile Leu Leu Ser Asp Ile Leu
370 375 380
Arg Val Asn Thr Glu Ile Thr Lys Ala Pro Leu Ser Ala Ser Met Ile
385 390 395 400
Lys Arg Tyr Asp Glu His His Gln Asp Leu Thr Leu Leu Lys Ala Leu
405 410 415
Val Arg Gln Gln Leu Pro Glu Lys Tyr Lys Glu Ile Phe Phe Asp Gln
420 425 430
Ser Lys Asn Gly Tyr Ala Gly Tyr Ile Asp Gly Gly Ala Ser Gln Glu
435 440 445
Glu Phe Tyr Lys Phe Ile Lys Pro Ile Leu Glu Lys Met Asp Gly Thr
450 455 460
Glu Glu Leu Leu Val Lys Leu Asn Arg Glu Asp Leu Leu Arg Lys Gln
465 470 475 480
Arg Thr Phe Asp Asn Gly Ser Ile Pro His Gln Ile His Leu Gly Glu
485 490 495
Leu His Ala Ile Leu Arg Arg Gln Glu Asp Phe Tyr Pro Phe Leu Lys
500 505 510
Asp Asn Arg Glu Lys Ile Glu Lys Ile Leu Thr Phe Arg Ile Pro Tyr
515 520 525
Tyr Val Gly Pro Leu Ala Arg Gly Asn Ser Arg Phe Ala Trp Met Thr
530 535 540
Arg Lys Ser Glu Glu Thr Ile Thr Pro Trp Asn Phe Glu Glu Val Val
545 550 555 560
Asp Lys Gly Ala Ser Ala Gln Ser Phe Ile Glu Arg Met Thr Asn Phe
565 570 575
Asp Lys Asn Leu Pro Asn Glu Lys Val Leu Pro Lys His Ser Leu Leu
580 585 590
Tyr Glu Tyr Phe Thr Val Tyr Asn Glu Leu Thr Lys Val Lys Tyr Val
595 600 605
Thr Glu Gly Met Arg Lys Pro Ala Phe Leu Ser Gly Glu Gln Lys Lys
610 615 620
Ala Ile Val Asp Leu Leu Phe Lys Thr Asn Arg Lys Val Thr Val Lys
625 630 635 640
Gln Leu Lys Glu Asp Tyr Phe Lys Lys Ile Glu Cys Phe Asp Ser Val
645 650 655
Glu Ile Ser Gly Val Glu Asp Arg Phe Asn Ala Ser Leu Gly Thr Tyr
660 665 670
His Asp Leu Leu Lys Ile Ile Lys Asp Lys Asp Phe Leu Asp Asn Glu
675 680 685
Glu Asn Glu Asp Ile Leu Glu Asp Ile Val Leu Thr Leu Thr Leu Phe
690 695 700
Glu Asp Arg Glu Met Ile Glu Glu Arg Leu Lys Thr Tyr Ala His Leu
705 710 715 720
Phe Asp Asp Lys Val Met Lys Gln Leu Lys Arg Arg Arg Tyr Thr Gly
725 730 735
Trp Gly Arg Leu Ser Arg Lys Leu Ile Asn Gly Ile Arg Asp Lys Gln
740 745 750
Ser Gly Lys Thr Ile Leu Asp Phe Leu Lys Ser Asp Gly Phe Ala Asn
755 760 765
Arg Asn Phe Met Gln Leu Ile His Asp Asp Ser Leu Thr Phe Lys Glu
770 775 780
Asp Ile Gln Lys Ala Gln Val Ser Gly Gln Gly Asp Ser Leu His Glu
785 790 795 800
His Ile Ala Asn Leu Ala Gly Ser Pro Ala Ile Lys Lys Gly Ile Leu
805 810 815
Gln Thr Val Lys Val Val Asp Glu Leu Val Lys Val Met Gly Arg His
820 825 830
Lys Pro Glu Asn Ile Val Ile Glu Met Ala Arg Glu Asn Gln Thr Thr
835 840 845
Gln Lys Gly Gln Lys Asn Ser Arg Glu Arg Met Lys Arg Ile Glu Glu
850 855 860
Gly Ile Lys Glu Leu Gly Ser Gln Ile Leu Lys Glu His Pro Val Glu
865 870 875 880
Asn Thr Gln Leu Gln Asn Glu Lys Leu Tyr Leu Tyr Tyr Leu Gln Asn
885 890 895
Gly Arg Asp Met Tyr Val Asp Gln Glu Leu Asp Ile Asn Arg Leu Ser
900 905 910
Asp Tyr Asp Val Asp Ala Ile Val Pro Gln Ser Phe Leu Lys Asp Asp
915 920 925
Ser Ile Asp Asn Lys Val Leu Thr Arg Ser Asp Lys Asn Arg Gly Lys
930 935 940
Ser Asp Asn Val Pro Ser Glu Glu Val Val Lys Lys Met Lys Asn Tyr
945 950 955 960
Trp Arg Gln Leu Leu Asn Ala Lys Leu Ile Thr Gln Arg Lys Phe Asp
965 970 975
Asn Leu Thr Lys Ala Glu Arg Gly Gly Leu Ser Glu Leu Asp Lys Ala
980 985 990
Gly Phe Ile Lys Arg Gln Leu Val Glu Thr Arg Gln Ile Thr Lys His
995 1000 1005
Val Ala Gln Ile Leu Asp Ser Arg Met Asn Thr Lys Tyr Asp Glu
1010 1015 1020
Asn Asp Lys Leu Ile Arg Glu Val Lys Val Ile Thr Leu Lys Ser
1025 1030 1035
Lys Leu Val Ser Asp Phe Arg Lys Asp Phe Gln Phe Tyr Lys Val
1040 1045 1050
Arg Glu Ile Asn Asn Tyr His His Ala His Asp Ala Tyr Leu Asn
1055 1060 1065
Ala Val Val Gly Thr Ala Leu Ile Lys Lys Tyr Pro Lys Leu Glu
1070 1075 1080
Ser Glu Phe Val Tyr Gly Asp Tyr Lys Val Tyr Asp Val Arg Lys
1085 1090 1095
Met Ile Ala Lys Ser Glu Gln Glu Ile Gly Lys Ala Thr Ala Lys
1100 1105 1110
Tyr Phe Phe Tyr Ser Asn Ile Met Asn Phe Phe Lys Thr Glu Ile
1115 1120 1125
Thr Leu Ala Asn Gly Glu Ile Arg Lys Arg Pro Leu Ile Glu Thr
1130 1135 1140
Asn Gly Glu Thr Gly Glu Ile Val Trp Asp Lys Gly Arg Asp Phe
1145 1150 1155
Ala Thr Val Arg Lys Val Leu Ser Met Pro Gln Val Asn Ile Val
1160 1165 1170
Lys Lys Thr Glu Val Gln Thr Gly Gly Phe Ser Lys Glu Ser Ile
1175 1180 1185
Leu Pro Lys Arg Asn Ser Asp Lys Leu Ile Ala Arg Lys Lys Asp
1190 1195 1200
Trp Asp Pro Lys Lys Tyr Gly Gly Phe Asp Ser Pro Thr Val Ala
1205 1210 1215
Tyr Ser Val Leu Val Val Ala Lys Val Glu Lys Gly Lys Ser Lys
1220 1225 1230
Lys Leu Lys Ser Val Lys Glu Leu Leu Gly Ile Thr Ile Met Glu
1235 1240 1245
Arg Ser Ser Phe Glu Lys Asn Pro Ile Asp Phe Leu Glu Ala Lys
1250 1255 1260
Gly Tyr Lys Glu Val Lys Lys Asp Leu Ile Ile Lys Leu Pro Lys
1265 1270 1275
Tyr Ser Leu Phe Glu Leu Glu Asn Gly Arg Lys Arg Met Leu Ala
1280 1285 1290
Ser Ala Gly Glu Leu Gln Lys Gly Asn Glu Leu Ala Leu Pro Ser
1295 1300 1305
Lys Tyr Val Asn Phe Leu Tyr Leu Ala Ser His Tyr Glu Lys Leu
1310 1315 1320
Lys Gly Ser Pro Glu Asp Asn Glu Gln Lys Gln Leu Phe Val Glu
1325 1330 1335
Gln His Lys His Tyr Leu Asp Glu Ile Ile Glu Gln Ile Ser Glu
1340 1345 1350
Phe Ser Lys Arg Val Ile Leu Ala Asp Ala Asn Leu Asp Lys Val
1355 1360 1365
Leu Ser Ala Tyr Asn Lys His Arg Asp Lys Pro Ile Arg Glu Gln
1370 1375 1380
Ala Glu Asn Ile Ile His Leu Phe Thr Leu Thr Asn Leu Gly Ala
1385 1390 1395
Pro Ala Ala Phe Lys Tyr Phe Asp Thr Thr Ile Asp Arg Lys Arg
1400 1405 1410
Tyr Thr Ser Thr Lys Glu Val Leu Asp Ala Thr Leu Ile His Gln
1415 1420 1425
Ser Ile Thr Gly Leu Tyr Glu Thr Arg Ile Asp Leu Ser Gln Leu
1430 1435 1440
Gly Gly Asp Ser Arg Ala Asp Tyr Pro Tyr Asp Val Pro Asp Tyr
1445 1450 1455
Ala Ser Gly Ser Pro Lys Lys Lys Arg Lys Val Ser Pro Gly Gly
1460 1465 1470
Gly Pro Ser Ser Gly Ala Pro Pro Pro Ser Gly Gly Ser Pro Ala
1475 1480 1485
Gly Ser Pro Thr Ser Thr Glu Glu Gly Thr Ser Glu Ser Ala Thr
1490 1495 1500
Pro Glu Ser Gly Pro Gly Thr Ser Thr Glu Pro Ser Glu Gly Ser
1505 1510 1515
Ala Pro Gly Ser Pro Ala Gly Ser Pro Thr Ser Thr Glu Glu Gly
1520 1525 1530
Thr Ser Thr Glu Pro Ser Glu Gly Ser Ala Pro Gly Thr Ser Thr
1535 1540 1545
Glu Pro Ser Glu Asn His Asp Gln Glu Phe Asp Pro Pro Lys Val
1550 1555 1560
Tyr Pro Pro Val Pro Ala Glu Lys Arg Lys Pro Ile Arg Val Leu
1565 1570 1575
Ser Leu Phe Asp Gly Ile Ala Thr Gly Leu Leu Val Leu Lys Asp
1580 1585 1590
Leu Gly Ile Gln Val Asp Arg Tyr Ile Ala Ser Glu Val Cys Glu
1595 1600 1605
Asp Ser Ile Thr Val Gly Met Val Arg His Gln Gly Lys Ile Met
1610 1615 1620
Tyr Val Gly Asp Val Arg Ser Val Thr Gln Lys His Ile Gln Glu
1625 1630 1635
Trp Gly Pro Phe Asp Leu Val Ile Gly Gly Ser Pro Cys Asn Asp
1640 1645 1650
Leu Ser Ile Val Asn Pro Ala Arg Lys Gly Leu Tyr Glu Gly Thr
1655 1660 1665
Gly Arg Leu Phe Phe Glu Phe Tyr Arg Leu Leu His Asp Ala Arg
1670 1675 1680
Pro Lys Glu Gly Asp Asp Arg Pro Phe Phe Trp Leu Phe Glu Asn
1685 1690 1695
Val Val Ala Met Gly Val Ser Asp Lys Arg Asp Ile Ser Arg Phe
1700 1705 1710
Leu Glu Ser Asn Pro Val Met Ile Asp Ala Lys Glu Val Ser Ala
1715 1720 1725
Ala His Arg Ala Arg Tyr Phe Trp Gly Asn Leu Pro Gly Met Asn
1730 1735 1740
Arg Pro Leu Ala Ser Thr Val Asn Asp Lys Leu Glu Leu Gln Glu
1745 1750 1755
Cys Leu Glu His Gly Arg Ile Ala Lys Phe Ser Lys Val Arg Thr
1760 1765 1770
Ile Thr Thr Arg Ser Asn Ser Ile Lys Gln Gly Lys Asp Gln His
1775 1780 1785
Phe Pro Val Phe Met Asn Glu Lys Glu Asp Ile Leu Trp Cys Thr
1790 1795 1800
Glu Met Glu Arg Val Phe Gly Phe Pro Val His Tyr Thr Asp Val
1805 1810 1815
Ser Asn Met Ser Arg Leu Ala Arg Gln Arg Leu Leu Gly Arg Ser
1820 1825 1830
Trp Ser Val Pro Val Ile Arg His Leu Phe Ala Pro Leu Lys Glu
1835 1840 1845
Tyr Phe Ala Cys Val Ser Ser Gly Asn Ser Asn Ala Asn Ser Arg
1850 1855 1860
Gly Pro Ser Phe Ser Ser Gly Leu Val Pro Leu Ser Leu Arg Gly
1865 1870 1875
Ser His Met Gly Pro Met Glu Ile Tyr Lys Thr Val Ser Ala Trp
1880 1885 1890
Lys Arg Gln Pro Val Arg Val Leu Ser Leu Phe Arg Asn Ile Asp
1895 1900 1905
Lys Val Leu Lys Ser Leu Gly Phe Leu Glu Ser Gly Ser Gly Ser
1910 1915 1920
Gly Gly Gly Thr Leu Lys Tyr Val Glu Asp Val Thr Asn Val Val
1925 1930 1935
Arg Arg Asp Val Glu Lys Trp Gly Pro Phe Asp Leu Val Tyr Gly
1940 1945 1950
Ser Thr Gln Pro Leu Gly Ser Ser Cys Asp Arg Cys Pro Gly Trp
1955 1960 1965
Tyr Met Phe Gln Phe His Arg Ile Leu Gln Tyr Ala Leu Pro Arg
1970 1975 1980
Gln Glu Ser Gln Arg Pro Phe Phe Trp Ile Phe Met Asp Asn Leu
1985 1990 1995
Leu Leu Thr Glu Asp Asp Gln Glu Thr Thr Thr Arg Phe Leu Gln
2000 2005 2010
Thr Glu Ala Val Thr Leu Gln Asp Val Arg Gly Arg Asp Tyr Gln
2015 2020 2025
Asn Ala Met Arg Val Trp Ser Asn Ile Pro Gly Leu Lys Ser Lys
2030 2035 2040
His Ala Pro Leu Thr Pro Lys Glu Glu Glu Tyr Leu Gln Ala Gln
2045 2050 2055
Val Arg Ser Arg Ser Lys Leu Asp Ala Pro Lys Val Asp Leu Leu
2060 2065 2070
Val Lys Asn Cys Leu Leu Pro Leu Arg Glu Tyr Phe Lys Tyr Phe
2075 2080 2085
Ser Gln Asn Ser Leu Pro Leu Ser Arg Ala Asp Pro Lys Lys Lys
2090 2095 2100
Arg Lys Val Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu Lys Gln
2105 2110 2115
Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Gly Ser Gly Ala Thr
2120 2125 2130
Asn Phe Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro
2135 2140 2145
Gly Pro Ser Glu Leu Ile Lys Glu Asn Met His Met Lys Leu Tyr
2150 2155 2160
Met Glu Gly Thr Val Asp Asn His His Phe Lys Cys Thr Ser Glu
2165 2170 2175
Gly Glu Gly Lys Pro Tyr Glu Gly Thr Gln Thr Met Arg Ile Lys
2180 2185 2190
Val Val Glu Gly Gly Pro Leu Pro Phe Ala Phe Asp Ile Leu Ala
2195 2200 2205
Thr Ser Phe Leu Tyr Gly Ser Lys Thr Phe Ile Asn His Thr Gln
2210 2215 2220
Gly Ile Pro Asp Phe Phe Lys Gln Ser Phe Pro Glu Gly Phe Thr
2225 2230 2235
Trp Glu Arg Val Thr Thr Tyr Glu Asp Gly Gly Val Leu Thr Ala
2240 2245 2250
Thr Gln Asp Thr Ser Leu Gln Asp Gly Cys Leu Ile Tyr Asn Val
2255 2260 2265
Lys Ile Arg Gly Val Asn Phe Thr Ser Asn Gly Pro Val Met Gln
2270 2275 2280
Lys Lys Thr Leu Gly Trp Glu Ala Phe Thr Glu Thr Leu Tyr Pro
2285 2290 2295
Ala Asp Gly Gly Leu Glu Gly Arg Asn Asp Met Ala Leu Lys Leu
2300 2305 2310
Val Gly Gly Ser His Leu Ile Ala Asn Ile Lys Thr Thr Tyr Arg
2315 2320 2325
Ser Lys Lys Pro Ala Lys Asn Leu Lys Met Pro Gly Val Tyr Tyr
2330 2335 2340
Val Asp Tyr Arg Leu Glu Arg Ile Lys Glu Ala Asn Asn Glu Thr
2345 2350 2355
Tyr Val Glu Gln His Glu Val Ala Val Ala Arg Tyr Cys Asp Leu
2360 2365 2370
Pro Ser Lys Leu Gly His Lys Leu Asn
2375 2380
<210> 7
<211> 2303
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетический полипептид
<400> 7
Asp Ala Lys Ser Leu Thr Ala Trp Ser Arg Thr Leu Val Thr Phe Lys
1. 5 10 15
Asp Val Phe Val Asp Phe Thr Arg Glu Glu Trp Lys Leu Leu Asp Thr
20 25 30
Ala Gln Gln Ile Val Tyr Arg Asn Val Met Leu Glu Asn Tyr Lys Asn
35 40 45
Leu Val Ser Leu Gly Tyr Gln Leu Thr Lys Pro Asp Val Ile Leu Arg
50 55 60
Leu Glu Lys Gly Glu Glu Pro Ser Gly Ser Glu Thr Pro Gly Thr Ser
65 70 75 80
Glu Ser Ala Thr Pro Glu Ser Met Asp Lys Lys Tyr Ser Ile Gly Leu
85 90 95
Ala Ile Gly Thr Asn Ser Val Gly Trp Ala Val Ile Thr Asp Glu Tyr
100 105 110
Lys Val Pro Ser Lys Lys Phe Lys Val Leu Gly Asn Thr Asp Arg His
115 120 125
Ser Ile Lys Lys Asn Leu Ile Gly Ala Leu Leu Phe Asp Ser Gly Glu
130 135 140
Thr Ala Glu Ala Thr Arg Leu Lys Arg Thr Ala Arg Arg Arg Tyr Thr
145 150 155 160
Arg Arg Lys Asn Arg Ile Cys Tyr Leu Gln Glu Ile Phe Ser Asn Glu
165 170 175
Met Ala Lys Val Asp Asp Ser Phe Phe His Arg Leu Glu Glu Ser Phe
180 185 190
Leu Val Glu Glu Asp Lys Lys His Glu Arg His Pro Ile Phe Gly Asn
195 200 205
Ile Val Asp Glu Val Ala Tyr His Glu Lys Tyr Pro Thr Ile Tyr His
210 215 220
Leu Arg Lys Lys Leu Val Asp Ser Thr Asp Lys Ala Asp Leu Arg Leu
225 230 235 240
Ile Tyr Leu Ala Leu Ala His Met Ile Lys Phe Arg Gly His Phe Leu
245 250 255
Ile Glu Gly Asp Leu Asn Pro Asp Asn Ser Asp Val Asp Lys Leu Phe
260 265 270
Ile Gln Leu Val Gln Thr Tyr Asn Gln Leu Phe Glu Glu Asn Pro Ile
275 280 285
Asn Ala Ser Gly Val Asp Ala Lys Ala Ile Leu Ser Ala Arg Leu Ser
290 295 300
Lys Ser Arg Arg Leu Glu Asn Leu Ile Ala Gln Leu Pro Gly Glu Lys
305 310 315 320
Lys Asn Gly Leu Phe Gly Asn Leu Ile Ala Leu Ser Leu Gly Leu Thr
325 330 335
Pro Asn Phe Lys Ser Asn Phe Asp Leu Ala Glu Asp Ala Lys Leu Gln
340 345 350
Leu Ser Lys Asp Thr Tyr Asp Asp Asp Leu Asp Asn Leu Leu Ala Gln
355 360 365
Ile Gly Asp Gln Tyr Ala Asp Leu Phe Leu Ala Ala Lys Asn Leu Ser
370 375 380
Asp Ala Ile Leu Leu Ser Asp Ile Leu Arg Val Asn Thr Glu Ile Thr
385 390 395 400
Lys Ala Pro Leu Ser Ala Ser Met Ile Lys Arg Tyr Asp Glu His His
405 410 415
Gln Asp Leu Thr Leu Leu Lys Ala Leu Val Arg Gln Gln Leu Pro Glu
420 425 430
Lys Tyr Lys Glu Ile Phe Phe Asp Gln Ser Lys Asn Gly Tyr Ala Gly
435 440 445
Tyr Ile Asp Gly Gly Ala Ser Gln Glu Glu Phe Tyr Lys Phe Ile Lys
450 455 460
Pro Ile Leu Glu Lys Met Asp Gly Thr Glu Glu Leu Leu Val Lys Leu
465 470 475 480
Asn Arg Glu Asp Leu Leu Arg Lys Gln Arg Thr Phe Asp Asn Gly Ser
485 490 495
Ile Pro His Gln Ile His Leu Gly Glu Leu His Ala Ile Leu Arg Arg
500 505 510
Gln Glu Asp Phe Tyr Pro Phe Leu Lys Asp Asn Arg Glu Lys Ile Glu
515 520 525
Lys Ile Leu Thr Phe Arg Ile Pro Tyr Tyr Val Gly Pro Leu Ala Arg
530 535 540
Gly Asn Ser Arg Phe Ala Trp Met Thr Arg Lys Ser Glu Glu Thr Ile
545 550 555 560
Thr Pro Trp Asn Phe Glu Glu Val Val Asp Lys Gly Ala Ser Ala Gln
565 570 575
Ser Phe Ile Glu Arg Met Thr Asn Phe Asp Lys Asn Leu Pro Asn Glu
580 585 590
Lys Val Leu Pro Lys His Ser Leu Leu Tyr Glu Tyr Phe Thr Val Tyr
595 600 605
Asn Glu Leu Thr Lys Val Lys Tyr Val Thr Glu Gly Met Arg Lys Pro
610 615 620
Ala Phe Leu Ser Gly Glu Gln Lys Lys Ala Ile Val Asp Leu Leu Phe
625 630 635 640
Lys Thr Asn Arg Lys Val Thr Val Lys Gln Leu Lys Glu Asp Tyr Phe
645 650 655
Lys Lys Ile Glu Cys Phe Asp Ser Val Glu Ile Ser Gly Val Glu Asp
660 665 670
Arg Phe Asn Ala Ser Leu Gly Thr Tyr His Asp Leu Leu Lys Ile Ile
675 680 685
Lys Asp Lys Asp Phe Leu Asp Asn Glu Glu Asn Glu Asp Ile Leu Glu
690 695 700
Asp Ile Val Leu Thr Leu Thr Leu Phe Glu Asp Arg Glu Met Ile Glu
705 710 715 720
Glu Arg Leu Lys Thr Tyr Ala His Leu Phe Asp Asp Lys Val Met Lys
725 730 735
Gln Leu Lys Arg Arg Arg Tyr Thr Gly Trp Gly Arg Leu Ser Arg Lys
740 745 750
Leu Ile Asn Gly Ile Arg Asp Lys Gln Ser Gly Lys Thr Ile Leu Asp
755 760 765
Phe Leu Lys Ser Asp Gly Phe Ala Asn Arg Asn Phe Met Gln Leu Ile
770 775 780
His Asp Asp Ser Leu Thr Phe Lys Glu Asp Ile Gln Lys Ala Gln Val
785 790 795 800
Ser Gly Gln Gly Asp Ser Leu His Glu His Ile Ala Asn Leu Ala Gly
805 810 815
Ser Pro Ala Ile Lys Lys Gly Ile Leu Gln Thr Val Lys Val Val Asp
820 825 830
Glu Leu Val Lys Val Met Gly Arg His Lys Pro Glu Asn Ile Val Ile
835 840 845
Glu Met Ala Arg Glu Asn Gln Thr Thr Gln Lys Gly Gln Lys Asn Ser
850 855 860
Arg Glu Arg Met Lys Arg Ile Glu Glu Gly Ile Lys Glu Leu Gly Ser
865 870 875 880
Gln Ile Leu Lys Glu His Pro Val Glu Asn Thr Gln Leu Gln Asn Glu
885 890 895
Lys Leu Tyr Leu Tyr Tyr Leu Gln Asn Gly Arg Asp Met Tyr Val Asp
900 905 910
Gln Glu Leu Asp Ile Asn Arg Leu Ser Asp Tyr Asp Val Asp Ala Ile
915 920 925
Val Pro Gln Ser Phe Leu Lys Asp Asp Ser Ile Asp Asn Lys Val Leu
930 935 940
Thr Arg Ser Asp Lys Asn Arg Gly Lys Ser Asp Asn Val Pro Ser Glu
945 950 955 960
Glu Val Val Lys Lys Met Lys Asn Tyr Trp Arg Gln Leu Leu Asn Ala
965 970 975
Lys Leu Ile Thr Gln Arg Lys Phe Asp Asn Leu Thr Lys Ala Glu Arg
980 985 990
Gly Gly Leu Ser Glu Leu Asp Lys Ala Gly Phe Ile Lys Arg Gln Leu
995 1000 1005
Val Glu Thr Arg Gln Ile Thr Lys His Val Ala Gln Ile Leu Asp
1010 1015 1020
Ser Arg Met Asn Thr Lys Tyr Asp Glu Asn Asp Lys Leu Ile Arg
1025 1030 1035
Glu Val Lys Val Ile Thr Leu Lys Ser Lys Leu Val Ser Asp Phe
1040 1045 1050
Arg Lys Asp Phe Gln Phe Tyr Lys Val Arg Glu Ile Asn Asn Tyr
1055 1060 1065
His His Ala His Asp Ala Tyr Leu Asn Ala Val Val Gly Thr Ala
1070 1075 1080
Leu Ile Lys Lys Tyr Pro Lys Leu Glu Ser Glu Phe Val Tyr Gly
1085 1090 1095
Asp Tyr Lys Val Tyr Asp Val Arg Lys Met Ile Ala Lys Ser Glu
1100 1105 1110
Gln Glu Ile Gly Lys Ala Thr Ala Lys Tyr Phe Phe Tyr Ser Asn
1115 1120 1125
Ile Met Asn Phe Phe Lys Thr Glu Ile Thr Leu Ala Asn Gly Glu
1130 1135 1140
Ile Arg Lys Arg Pro Leu Ile Glu Thr Asn Gly Glu Thr Gly Glu
1145 1150 1155
Ile Val Trp Asp Lys Gly Arg Asp Phe Ala Thr Val Arg Lys Val
1160 1165 1170
Leu Ser Met Pro Gln Val Asn Ile Val Lys Lys Thr Glu Val Gln
1175 1180 1185
Thr Gly Gly Phe Ser Lys Glu Ser Ile Leu Pro Lys Arg Asn Ser
1190 1195 1200
Asp Lys Leu Ile Ala Arg Lys Lys Asp Trp Asp Pro Lys Lys Tyr
1205 1210 1215
Gly Gly Phe Asp Ser Pro Thr Val Ala Tyr Ser Val Leu Val Val
1220 1225 1230
Ala Lys Val Glu Lys Gly Lys Ser Lys Lys Leu Lys Ser Val Lys
1235 1240 1245
Glu Leu Leu Gly Ile Thr Ile Met Glu Arg Ser Ser Phe Glu Lys
1250 1255 1260
Asn Pro Ile Asp Phe Leu Glu Ala Lys Gly Tyr Lys Glu Val Lys
1265 1270 1275
Lys Asp Leu Ile Ile Lys Leu Pro Lys Tyr Ser Leu Phe Glu Leu
1280 1285 1290
Glu Asn Gly Arg Lys Arg Met Leu Ala Ser Ala Gly Glu Leu Gln
1295 1300 1305
Lys Gly Asn Glu Leu Ala Leu Pro Ser Lys Tyr Val Asn Phe Leu
1310 1315 1320
Tyr Leu Ala Ser His Tyr Glu Lys Leu Lys Gly Ser Pro Glu Asp
1325 1330 1335
Asn Glu Gln Lys Gln Leu Phe Val Glu Gln His Lys His Tyr Leu
1340 1345 1350
Asp Glu Ile Ile Glu Gln Ile Ser Glu Phe Ser Lys Arg Val Ile
1355 1360 1365
Leu Ala Asp Ala Asn Leu Asp Lys Val Leu Ser Ala Tyr Asn Lys
1370 1375 1380
His Arg Asp Lys Pro Ile Arg Glu Gln Ala Glu Asn Ile Ile His
1385 1390 1395
Leu Phe Thr Leu Thr Asn Leu Gly Ala Pro Ala Ala Phe Lys Tyr
1400 1405 1410
Phe Asp Thr Thr Ile Asp Arg Lys Arg Tyr Thr Ser Thr Lys Glu
1415 1420 1425
Val Leu Asp Ala Thr Leu Ile His Gln Ser Ile Thr Gly Leu Tyr
1430 1435 1440
Glu Thr Arg Ile Asp Leu Ser Gln Leu Gly Gly Asp Ser Arg Ala
1445 1450 1455
Asp Tyr Pro Tyr Asp Val Pro Asp Tyr Ala Ser Gly Ser Pro Lys
1460 1465 1470
Lys Lys Arg Lys Val Glu Ala Ser Gly Ser Gly Arg Ala Ser Pro
1475 1480 1485
Gly Ile Pro Gly Ser Thr Arg Asn His Asp Gln Glu Phe Asp Pro
1490 1495 1500
Pro Lys Val Tyr Pro Pro Val Pro Ala Glu Lys Arg Lys Pro Ile
1505 1510 1515
Arg Val Leu Ser Leu Phe Asp Gly Ile Ala Thr Gly Leu Leu Val
1520 1525 1530
Leu Lys Asp Leu Gly Ile Gln Val Asp Arg Tyr Ile Ala Ser Glu
1535 1540 1545
Val Cys Glu Asp Ser Ile Thr Val Gly Met Val Arg His Gln Gly
1550 1555 1560
Lys Ile Met Tyr Val Gly Asp Val Arg Ser Val Thr Gln Lys His
1565 1570 1575
Ile Gln Glu Trp Gly Pro Phe Asp Leu Val Ile Gly Gly Ser Pro
1580 1585 1590
Cys Asn Asp Leu Ser Ile Val Asn Pro Ala Arg Lys Gly Leu Tyr
1595 1600 1605
Glu Gly Thr Gly Arg Leu Phe Phe Glu Phe Tyr Arg Leu Leu His
1610 1615 1620
Asp Ala Arg Pro Lys Glu Gly Asp Asp Arg Pro Phe Phe Trp Leu
1625 1630 1635
Phe Glu Asn Val Val Ala Met Gly Val Ser Asp Lys Arg Asp Ile
1640 1645 1650
Ser Arg Phe Leu Glu Ser Asn Pro Val Met Ile Asp Ala Lys Glu
1655 1660 1665
Val Ser Ala Ala His Arg Ala Arg Tyr Phe Trp Gly Asn Leu Pro
1670 1675 1680
Gly Met Asn Arg Pro Leu Ala Ser Thr Val Asn Asp Lys Leu Glu
1685 1690 1695
Leu Gln Glu Cys Leu Glu His Gly Arg Ile Ala Lys Phe Ser Lys
1700 1705 1710
Val Arg Thr Ile Thr Thr Arg Ser Asn Ser Ile Lys Gln Gly Lys
1715 1720 1725
Asp Gln His Phe Pro Val Phe Met Asn Glu Lys Glu Asp Ile Leu
1730 1735 1740
Trp Cys Thr Glu Met Glu Arg Val Phe Gly Phe Pro Val His Tyr
1745 1750 1755
Thr Asp Val Ser Asn Met Ser Arg Leu Ala Arg Gln Arg Leu Leu
1760 1765 1770
Gly Arg Ser Trp Ser Val Pro Val Ile Arg His Leu Phe Ala Pro
1775 1780 1785
Leu Lys Glu Tyr Phe Ala Cys Val Ser Ser Gly Asn Ser Asn Ala
1790 1795 1800
Asn Ser Arg Gly Pro Ser Phe Ser Ser Gly Leu Val Pro Leu Ser
1805 1810 1815
Leu Arg Gly Ser His Met Gly Pro Met Glu Ile Tyr Lys Thr Val
1820 1825 1830
Ser Ala Trp Lys Arg Gln Pro Val Arg Val Leu Ser Leu Phe Arg
1835 1840 1845
Asn Ile Asp Lys Val Leu Lys Ser Leu Gly Phe Leu Glu Ser Gly
1850 1855 1860
Ser Gly Ser Gly Gly Gly Thr Leu Lys Tyr Val Glu Asp Val Thr
1865 1870 1875
Asn Val Val Arg Arg Asp Val Glu Lys Trp Gly Pro Phe Asp Leu
1880 1885 1890
Val Tyr Gly Ser Thr Gln Pro Leu Gly Ser Ser Cys Asp Arg Cys
1895 1900 1905
Pro Gly Trp Tyr Met Phe Gln Phe His Arg Ile Leu Gln Tyr Ala
1910 1915 1920
Leu Pro Arg Gln Glu Ser Gln Arg Pro Phe Phe Trp Ile Phe Met
1925 1930 1935
Asp Asn Leu Leu Leu Thr Glu Asp Asp Gln Glu Thr Thr Thr Arg
1940 1945 1950
Phe Leu Gln Thr Glu Ala Val Thr Leu Gln Asp Val Arg Gly Arg
1955 1960 1965
Asp Tyr Gln Asn Ala Met Arg Val Trp Ser Asn Ile Pro Gly Leu
1970 1975 1980
Lys Ser Lys His Ala Pro Leu Thr Pro Lys Glu Glu Glu Tyr Leu
1985 1990 1995
Gln Ala Gln Val Arg Ser Arg Ser Lys Leu Asp Ala Pro Lys Val
2000 2005 2010
Asp Leu Leu Val Lys Asn Cys Leu Leu Pro Leu Arg Glu Tyr Phe
2015 2020 2025
Lys Tyr Phe Ser Gln Asn Ser Leu Pro Leu Ser Arg Ala Asp Pro
2030 2035 2040
Lys Lys Lys Arg Lys Val Gly Ser Gly Ala Thr Asn Phe Ser Leu
2045 2050 2055
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Ser Glu
2060 2065 2070
Leu Ile Lys Glu Asn Met His Met Lys Leu Tyr Met Glu Gly Thr
2075 2080 2085
Val Asp Asn His His Phe Lys Cys Thr Ser Glu Gly Glu Gly Lys
2090 2095 2100
Pro Tyr Glu Gly Thr Gln Thr Met Arg Ile Lys Val Val Glu Gly
2105 2110 2115
Gly Pro Leu Pro Phe Ala Phe Asp Ile Leu Ala Thr Ser Phe Leu
2120 2125 2130
Tyr Gly Ser Lys Thr Phe Ile Asn His Thr Gln Gly Ile Pro Asp
2135 2140 2145
Phe Phe Lys Gln Ser Phe Pro Glu Gly Phe Thr Trp Glu Arg Val
2150 2155 2160
Thr Thr Tyr Glu Asp Gly Gly Val Leu Thr Ala Thr Gln Asp Thr
2165 2170 2175
Ser Leu Gln Asp Gly Cys Leu Ile Tyr Asn Val Lys Ile Arg Gly
2180 2185 2190
Val Asn Phe Thr Ser Asn Gly Pro Val Met Gln Lys Lys Thr Leu
2195 2200 2205
Gly Trp Glu Ala Phe Thr Glu Thr Leu Tyr Pro Ala Asp Gly Gly
2210 2215 2220
Leu Glu Gly Arg Asn Asp Met Ala Leu Lys Leu Val Gly Gly Ser
2225 2230 2235
His Leu Ile Ala Asn Ile Lys Thr Thr Tyr Arg Ser Lys Lys Pro
2240 2245 2250
Ala Lys Asn Leu Lys Met Pro Gly Val Tyr Tyr Val Asp Tyr Arg
2255 2260 2265
Leu Glu Arg Ile Lys Glu Ala Asn Asn Glu Thr Tyr Val Glu Gln
2270 2275 2280
His Glu Val Ala Val Ala Arg Tyr Cys Asp Leu Pro Ser Lys Leu
2285 2290 2295
Gly His Lys Leu Asn
2300
<210> 8
<211> 2325
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетический полипептид
<400> 8
Asp Ala Lys Ser Leu Thr Ala Trp Ser Arg Thr Leu Val Thr Phe Lys
1. 5 10 15
Asp Val Phe Val Asp Phe Thr Arg Glu Glu Trp Lys Leu Leu Asp Thr
20 25 30
Ala Gln Gln Ile Val Tyr Arg Asn Val Met Leu Glu Asn Tyr Lys Asn
35 40 45
Leu Val Ser Leu Gly Tyr Gln Leu Thr Lys Pro Asp Val Ile Leu Arg
50 55 60
Leu Glu Lys Gly Glu Glu Pro Ser Gly Ser Glu Thr Pro Gly Thr Ser
65 70 75 80
Glu Ser Ala Thr Pro Glu Ser Met Asp Lys Lys Tyr Ser Ile Gly Leu
85 90 95
Ala Ile Gly Thr Asn Ser Val Gly Trp Ala Val Ile Thr Asp Glu Tyr
100 105 110
Lys Val Pro Ser Lys Lys Phe Lys Val Leu Gly Asn Thr Asp Arg His
115 120 125
Ser Ile Lys Lys Asn Leu Ile Gly Ala Leu Leu Phe Asp Ser Gly Glu
130 135 140
Thr Ala Glu Ala Thr Arg Leu Lys Arg Thr Ala Arg Arg Arg Tyr Thr
145 150 155 160
Arg Arg Lys Asn Arg Ile Cys Tyr Leu Gln Glu Ile Phe Ser Asn Glu
165 170 175
Met Ala Lys Val Asp Asp Ser Phe Phe His Arg Leu Glu Glu Ser Phe
180 185 190
Leu Val Glu Glu Asp Lys Lys His Glu Arg His Pro Ile Phe Gly Asn
195 200 205
Ile Val Asp Glu Val Ala Tyr His Glu Lys Tyr Pro Thr Ile Tyr His
210 215 220
Leu Arg Lys Lys Leu Val Asp Ser Thr Asp Lys Ala Asp Leu Arg Leu
225 230 235 240
Ile Tyr Leu Ala Leu Ala His Met Ile Lys Phe Arg Gly His Phe Leu
245 250 255
Ile Glu Gly Asp Leu Asn Pro Asp Asn Ser Asp Val Asp Lys Leu Phe
260 265 270
Ile Gln Leu Val Gln Thr Tyr Asn Gln Leu Phe Glu Glu Asn Pro Ile
275 280 285
Asn Ala Ser Gly Val Asp Ala Lys Ala Ile Leu Ser Ala Arg Leu Ser
290 295 300
Lys Ser Arg Arg Leu Glu Asn Leu Ile Ala Gln Leu Pro Gly Glu Lys
305 310 315 320
Lys Asn Gly Leu Phe Gly Asn Leu Ile Ala Leu Ser Leu Gly Leu Thr
325 330 335
Pro Asn Phe Lys Ser Asn Phe Asp Leu Ala Glu Asp Ala Lys Leu Gln
340 345 350
Leu Ser Lys Asp Thr Tyr Asp Asp Asp Leu Asp Asn Leu Leu Ala Gln
355 360 365
Ile Gly Asp Gln Tyr Ala Asp Leu Phe Leu Ala Ala Lys Asn Leu Ser
370 375 380
Asp Ala Ile Leu Leu Ser Asp Ile Leu Arg Val Asn Thr Glu Ile Thr
385 390 395 400
Lys Ala Pro Leu Ser Ala Ser Met Ile Lys Arg Tyr Asp Glu His His
405 410 415
Gln Asp Leu Thr Leu Leu Lys Ala Leu Val Arg Gln Gln Leu Pro Glu
420 425 430
Lys Tyr Lys Glu Ile Phe Phe Asp Gln Ser Lys Asn Gly Tyr Ala Gly
435 440 445
Tyr Ile Asp Gly Gly Ala Ser Gln Glu Glu Phe Tyr Lys Phe Ile Lys
450 455 460
Pro Ile Leu Glu Lys Met Asp Gly Thr Glu Glu Leu Leu Val Lys Leu
465 470 475 480
Asn Arg Glu Asp Leu Leu Arg Lys Gln Arg Thr Phe Asp Asn Gly Ser
485 490 495
Ile Pro His Gln Ile His Leu Gly Glu Leu His Ala Ile Leu Arg Arg
500 505 510
Gln Glu Asp Phe Tyr Pro Phe Leu Lys Asp Asn Arg Glu Lys Ile Glu
515 520 525
Lys Ile Leu Thr Phe Arg Ile Pro Tyr Tyr Val Gly Pro Leu Ala Arg
530 535 540
Gly Asn Ser Arg Phe Ala Trp Met Thr Arg Lys Ser Glu Glu Thr Ile
545 550 555 560
Thr Pro Trp Asn Phe Glu Glu Val Val Asp Lys Gly Ala Ser Ala Gln
565 570 575
Ser Phe Ile Glu Arg Met Thr Asn Phe Asp Lys Asn Leu Pro Asn Glu
580 585 590
Lys Val Leu Pro Lys His Ser Leu Leu Tyr Glu Tyr Phe Thr Val Tyr
595 600 605
Asn Glu Leu Thr Lys Val Lys Tyr Val Thr Glu Gly Met Arg Lys Pro
610 615 620
Ala Phe Leu Ser Gly Glu Gln Lys Lys Ala Ile Val Asp Leu Leu Phe
625 630 635 640
Lys Thr Asn Arg Lys Val Thr Val Lys Gln Leu Lys Glu Asp Tyr Phe
645 650 655
Lys Lys Ile Glu Cys Phe Asp Ser Val Glu Ile Ser Gly Val Glu Asp
660 665 670
Arg Phe Asn Ala Ser Leu Gly Thr Tyr His Asp Leu Leu Lys Ile Ile
675 680 685
Lys Asp Lys Asp Phe Leu Asp Asn Glu Glu Asn Glu Asp Ile Leu Glu
690 695 700
Asp Ile Val Leu Thr Leu Thr Leu Phe Glu Asp Arg Glu Met Ile Glu
705 710 715 720
Glu Arg Leu Lys Thr Tyr Ala His Leu Phe Asp Asp Lys Val Met Lys
725 730 735
Gln Leu Lys Arg Arg Arg Tyr Thr Gly Trp Gly Arg Leu Ser Arg Lys
740 745 750
Leu Ile Asn Gly Ile Arg Asp Lys Gln Ser Gly Lys Thr Ile Leu Asp
755 760 765
Phe Leu Lys Ser Asp Gly Phe Ala Asn Arg Asn Phe Met Gln Leu Ile
770 775 780
His Asp Asp Ser Leu Thr Phe Lys Glu Asp Ile Gln Lys Ala Gln Val
785 790 795 800
Ser Gly Gln Gly Asp Ser Leu His Glu His Ile Ala Asn Leu Ala Gly
805 810 815
Ser Pro Ala Ile Lys Lys Gly Ile Leu Gln Thr Val Lys Val Val Asp
820 825 830
Glu Leu Val Lys Val Met Gly Arg His Lys Pro Glu Asn Ile Val Ile
835 840 845
Glu Met Ala Arg Glu Asn Gln Thr Thr Gln Lys Gly Gln Lys Asn Ser
850 855 860
Arg Glu Arg Met Lys Arg Ile Glu Glu Gly Ile Lys Glu Leu Gly Ser
865 870 875 880
Gln Ile Leu Lys Glu His Pro Val Glu Asn Thr Gln Leu Gln Asn Glu
885 890 895
Lys Leu Tyr Leu Tyr Tyr Leu Gln Asn Gly Arg Asp Met Tyr Val Asp
900 905 910
Gln Glu Leu Asp Ile Asn Arg Leu Ser Asp Tyr Asp Val Asp Ala Ile
915 920 925
Val Pro Gln Ser Phe Leu Lys Asp Asp Ser Ile Asp Asn Lys Val Leu
930 935 940
Thr Arg Ser Asp Lys Asn Arg Gly Lys Ser Asp Asn Val Pro Ser Glu
945 950 955 960
Glu Val Val Lys Lys Met Lys Asn Tyr Trp Arg Gln Leu Leu Asn Ala
965 970 975
Lys Leu Ile Thr Gln Arg Lys Phe Asp Asn Leu Thr Lys Ala Glu Arg
980 985 990
Gly Gly Leu Ser Glu Leu Asp Lys Ala Gly Phe Ile Lys Arg Gln Leu
995 1000 1005
Val Glu Thr Arg Gln Ile Thr Lys His Val Ala Gln Ile Leu Asp
1010 1015 1020
Ser Arg Met Asn Thr Lys Tyr Asp Glu Asn Asp Lys Leu Ile Arg
1025 1030 1035
Glu Val Lys Val Ile Thr Leu Lys Ser Lys Leu Val Ser Asp Phe
1040 1045 1050
Arg Lys Asp Phe Gln Phe Tyr Lys Val Arg Glu Ile Asn Asn Tyr
1055 1060 1065
His His Ala His Asp Ala Tyr Leu Asn Ala Val Val Gly Thr Ala
1070 1075 1080
Leu Ile Lys Lys Tyr Pro Lys Leu Glu Ser Glu Phe Val Tyr Gly
1085 1090 1095
Asp Tyr Lys Val Tyr Asp Val Arg Lys Met Ile Ala Lys Ser Glu
1100 1105 1110
Gln Glu Ile Gly Lys Ala Thr Ala Lys Tyr Phe Phe Tyr Ser Asn
1115 1120 1125
Ile Met Asn Phe Phe Lys Thr Glu Ile Thr Leu Ala Asn Gly Glu
1130 1135 1140
Ile Arg Lys Arg Pro Leu Ile Glu Thr Asn Gly Glu Thr Gly Glu
1145 1150 1155
Ile Val Trp Asp Lys Gly Arg Asp Phe Ala Thr Val Arg Lys Val
1160 1165 1170
Leu Ser Met Pro Gln Val Asn Ile Val Lys Lys Thr Glu Val Gln
1175 1180 1185
Thr Gly Gly Phe Ser Lys Glu Ser Ile Leu Pro Lys Arg Asn Ser
1190 1195 1200
Asp Lys Leu Ile Ala Arg Lys Lys Asp Trp Asp Pro Lys Lys Tyr
1205 1210 1215
Gly Gly Phe Asp Ser Pro Thr Val Ala Tyr Ser Val Leu Val Val
1220 1225 1230
Ala Lys Val Glu Lys Gly Lys Ser Lys Lys Leu Lys Ser Val Lys
1235 1240 1245
Glu Leu Leu Gly Ile Thr Ile Met Glu Arg Ser Ser Phe Glu Lys
1250 1255 1260
Asn Pro Ile Asp Phe Leu Glu Ala Lys Gly Tyr Lys Glu Val Lys
1265 1270 1275
Lys Asp Leu Ile Ile Lys Leu Pro Lys Tyr Ser Leu Phe Glu Leu
1280 1285 1290
Glu Asn Gly Arg Lys Arg Met Leu Ala Ser Ala Gly Glu Leu Gln
1295 1300 1305
Lys Gly Asn Glu Leu Ala Leu Pro Ser Lys Tyr Val Asn Phe Leu
1310 1315 1320
Tyr Leu Ala Ser His Tyr Glu Lys Leu Lys Gly Ser Pro Glu Asp
1325 1330 1335
Asn Glu Gln Lys Gln Leu Phe Val Glu Gln His Lys His Tyr Leu
1340 1345 1350
Asp Glu Ile Ile Glu Gln Ile Ser Glu Phe Ser Lys Arg Val Ile
1355 1360 1365
Leu Ala Asp Ala Asn Leu Asp Lys Val Leu Ser Ala Tyr Asn Lys
1370 1375 1380
His Arg Asp Lys Pro Ile Arg Glu Gln Ala Glu Asn Ile Ile His
1385 1390 1395
Leu Phe Thr Leu Thr Asn Leu Gly Ala Pro Ala Ala Phe Lys Tyr
1400 1405 1410
Phe Asp Thr Thr Ile Asp Arg Lys Arg Tyr Thr Ser Thr Lys Glu
1415 1420 1425
Val Leu Asp Ala Thr Leu Ile His Gln Ser Ile Thr Gly Leu Tyr
1430 1435 1440
Glu Thr Arg Ile Asp Leu Ser Gln Leu Gly Gly Asp Ser Arg Ala
1445 1450 1455
Asp Tyr Pro Tyr Asp Val Pro Asp Tyr Ala Ser Gly Ser Pro Lys
1460 1465 1470
Lys Lys Arg Lys Val Glu Ala Ser Gly Ser Gly Arg Ala Ser Pro
1475 1480 1485
Gly Ile Pro Gly Ser Thr Arg Asn His Asp Gln Glu Phe Asp Pro
1490 1495 1500
Pro Lys Val Tyr Pro Pro Val Pro Ala Glu Lys Arg Lys Pro Ile
1505 1510 1515
Arg Val Leu Ser Leu Phe Asp Gly Ile Ala Thr Gly Leu Leu Val
1520 1525 1530
Leu Lys Asp Leu Gly Ile Gln Val Asp Arg Tyr Ile Ala Ser Glu
1535 1540 1545
Val Cys Glu Asp Ser Ile Thr Val Gly Met Val Arg His Gln Gly
1550 1555 1560
Lys Ile Met Tyr Val Gly Asp Val Arg Ser Val Thr Gln Lys His
1565 1570 1575
Ile Gln Glu Trp Gly Pro Phe Asp Leu Val Ile Gly Gly Ser Pro
1580 1585 1590
Cys Asn Asp Leu Ser Ile Val Asn Pro Ala Arg Lys Gly Leu Tyr
1595 1600 1605
Glu Gly Thr Gly Arg Leu Phe Phe Glu Phe Tyr Arg Leu Leu His
1610 1615 1620
Asp Ala Arg Pro Lys Glu Gly Asp Asp Arg Pro Phe Phe Trp Leu
1625 1630 1635
Phe Glu Asn Val Val Ala Met Gly Val Ser Asp Lys Arg Asp Ile
1640 1645 1650
Ser Arg Phe Leu Glu Ser Asn Pro Val Met Ile Asp Ala Lys Glu
1655 1660 1665
Val Ser Ala Ala His Arg Ala Arg Tyr Phe Trp Gly Asn Leu Pro
1670 1675 1680
Gly Met Asn Arg Pro Leu Ala Ser Thr Val Asn Asp Lys Leu Glu
1685 1690 1695
Leu Gln Glu Cys Leu Glu His Gly Arg Ile Ala Lys Phe Ser Lys
1700 1705 1710
Val Arg Thr Ile Thr Thr Arg Ser Asn Ser Ile Lys Gln Gly Lys
1715 1720 1725
Asp Gln His Phe Pro Val Phe Met Asn Glu Lys Glu Asp Ile Leu
1730 1735 1740
Trp Cys Thr Glu Met Glu Arg Val Phe Gly Phe Pro Val His Tyr
1745 1750 1755
Thr Asp Val Ser Asn Met Ser Arg Leu Ala Arg Gln Arg Leu Leu
1760 1765 1770
Gly Arg Ser Trp Ser Val Pro Val Ile Arg His Leu Phe Ala Pro
1775 1780 1785
Leu Lys Glu Tyr Phe Ala Cys Val Ser Ser Gly Asn Ser Asn Ala
1790 1795 1800
Asn Ser Arg Gly Pro Ser Phe Ser Ser Gly Leu Val Pro Leu Ser
1805 1810 1815
Leu Arg Gly Ser His Met Gly Pro Met Glu Ile Tyr Lys Thr Val
1820 1825 1830
Ser Ala Trp Lys Arg Gln Pro Val Arg Val Leu Ser Leu Phe Arg
1835 1840 1845
Asn Ile Asp Lys Val Leu Lys Ser Leu Gly Phe Leu Glu Ser Gly
1850 1855 1860
Ser Gly Ser Gly Gly Gly Thr Leu Lys Tyr Val Glu Asp Val Thr
1865 1870 1875
Asn Val Val Arg Arg Asp Val Glu Lys Trp Gly Pro Phe Asp Leu
1880 1885 1890
Val Tyr Gly Ser Thr Gln Pro Leu Gly Ser Ser Cys Asp Arg Cys
1895 1900 1905
Pro Gly Trp Tyr Met Phe Gln Phe His Arg Ile Leu Gln Tyr Ala
1910 1915 1920
Leu Pro Arg Gln Glu Ser Gln Arg Pro Phe Phe Trp Ile Phe Met
1925 1930 1935
Asp Asn Leu Leu Leu Thr Glu Asp Asp Gln Glu Thr Thr Thr Arg
1940 1945 1950
Phe Leu Gln Thr Glu Ala Val Thr Leu Gln Asp Val Arg Gly Arg
1955 1960 1965
Asp Tyr Gln Asn Ala Met Arg Val Trp Ser Asn Ile Pro Gly Leu
1970 1975 1980
Lys Ser Lys His Ala Pro Leu Thr Pro Lys Glu Glu Glu Tyr Leu
1985 1990 1995
Gln Ala Gln Val Arg Ser Arg Ser Lys Leu Asp Ala Pro Lys Val
2000 2005 2010
Asp Leu Leu Val Lys Asn Cys Leu Leu Pro Leu Arg Glu Tyr Phe
2015 2020 2025
Lys Tyr Phe Ser Gln Asn Ser Leu Pro Leu Ser Arg Ala Asp Pro
2030 2035 2040
Lys Lys Lys Arg Lys Val Gly Ser Gly Ala Thr Asn Phe Ser Leu
2045 2050 2055
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Gly Ser
2060 2065 2070
Gly Ala Thr Asn Phe Ser Leu Leu Lys Gln Ala Gly Asp Val Glu
2075 2080 2085
Glu Asn Pro Gly Pro Ser Glu Leu Ile Lys Glu Asn Met His Met
2090 2095 2100
Lys Leu Tyr Met Glu Gly Thr Val Asp Asn His His Phe Lys Cys
2105 2110 2115
Thr Ser Glu Gly Glu Gly Lys Pro Tyr Glu Gly Thr Gln Thr Met
2120 2125 2130
Arg Ile Lys Val Val Glu Gly Gly Pro Leu Pro Phe Ala Phe Asp
2135 2140 2145
Ile Leu Ala Thr Ser Phe Leu Tyr Gly Ser Lys Thr Phe Ile Asn
2150 2155 2160
His Thr Gln Gly Ile Pro Asp Phe Phe Lys Gln Ser Phe Pro Glu
2165 2170 2175
Gly Phe Thr Trp Glu Arg Val Thr Thr Tyr Glu Asp Gly Gly Val
2180 2185 2190
Leu Thr Ala Thr Gln Asp Thr Ser Leu Gln Asp Gly Cys Leu Ile
2195 2200 2205
Tyr Asn Val Lys Ile Arg Gly Val Asn Phe Thr Ser Asn Gly Pro
2210 2215 2220
Val Met Gln Lys Lys Thr Leu Gly Trp Glu Ala Phe Thr Glu Thr
2225 2230 2235
Leu Tyr Pro Ala Asp Gly Gly Leu Glu Gly Arg Asn Asp Met Ala
2240 2245 2250
Leu Lys Leu Val Gly Gly Ser His Leu Ile Ala Asn Ile Lys Thr
2255 2260 2265
Thr Tyr Arg Ser Lys Lys Pro Ala Lys Asn Leu Lys Met Pro Gly
2270 2275 2280
Val Tyr Tyr Val Asp Tyr Arg Leu Glu Arg Ile Lys Glu Ala Asn
2285 2290 2295
Asn Glu Thr Tyr Val Glu Gln His Glu Val Ala Val Ala Arg Tyr
2300 2305 2310
Cys Asp Leu Pro Ser Lys Leu Gly His Lys Leu Asn
2315 2320 2325
<210> 9
<211> 2367
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетический полипептид
<400> 9
Asp Ala Lys Ser Leu Thr Ala Trp Ser Arg Thr Leu Val Thr Phe Lys
1. 5 10 15
Asp Val Phe Val Asp Phe Thr Arg Glu Glu Trp Lys Leu Leu Asp Thr
20 25 30
Ala Gln Gln Ile Val Tyr Arg Asn Val Met Leu Glu Asn Tyr Lys Asn
35 40 45
Leu Val Ser Leu Gly Tyr Gln Leu Thr Lys Pro Asp Val Ile Leu Arg
50 55 60
Leu Glu Lys Gly Glu Glu Pro Gly Gly Pro Ser Ser Gly Ala Pro Pro
65 70 75 80
Pro Ser Gly Gly Ser Pro Ala Gly Ser Pro Thr Ser Thr Glu Glu Gly
85 90 95
Thr Ser Glu Ser Ala Thr Pro Glu Ser Gly Pro Gly Thr Ser Thr Glu
100 105 110
Pro Ser Glu Gly Ser Ala Pro Gly Ser Pro Ala Gly Ser Pro Thr Ser
115 120 125
Thr Glu Glu Gly Thr Ser Thr Glu Pro Ser Glu Gly Ser Ala Pro Gly
130 135 140
Thr Ser Thr Glu Pro Ser Glu Met Asp Lys Lys Tyr Ser Ile Gly Leu
145 150 155 160
Ala Ile Gly Thr Asn Ser Val Gly Trp Ala Val Ile Thr Asp Glu Tyr
165 170 175
Lys Val Pro Ser Lys Lys Phe Lys Val Leu Gly Asn Thr Asp Arg His
180 185 190
Ser Ile Lys Lys Asn Leu Ile Gly Ala Leu Leu Phe Asp Ser Gly Glu
195 200 205
Thr Ala Glu Ala Thr Arg Leu Lys Arg Thr Ala Arg Arg Arg Tyr Thr
210 215 220
Arg Arg Lys Asn Arg Ile Cys Tyr Leu Gln Glu Ile Phe Ser Asn Glu
225 230 235 240
Met Ala Lys Val Asp Asp Ser Phe Phe His Arg Leu Glu Glu Ser Phe
245 250 255
Leu Val Glu Glu Asp Lys Lys His Glu Arg His Pro Ile Phe Gly Asn
260 265 270
Ile Val Asp Glu Val Ala Tyr His Glu Lys Tyr Pro Thr Ile Tyr His
275 280 285
Leu Arg Lys Lys Leu Val Asp Ser Thr Asp Lys Ala Asp Leu Arg Leu
290 295 300
Ile Tyr Leu Ala Leu Ala His Met Ile Lys Phe Arg Gly His Phe Leu
305 310 315 320
Ile Glu Gly Asp Leu Asn Pro Asp Asn Ser Asp Val Asp Lys Leu Phe
325 330 335
Ile Gln Leu Val Gln Thr Tyr Asn Gln Leu Phe Glu Glu Asn Pro Ile
340 345 350
Asn Ala Ser Gly Val Asp Ala Lys Ala Ile Leu Ser Ala Arg Leu Ser
355 360 365
Lys Ser Arg Arg Leu Glu Asn Leu Ile Ala Gln Leu Pro Gly Glu Lys
370 375 380
Lys Asn Gly Leu Phe Gly Asn Leu Ile Ala Leu Ser Leu Gly Leu Thr
385 390 395 400
Pro Asn Phe Lys Ser Asn Phe Asp Leu Ala Glu Asp Ala Lys Leu Gln
405 410 415
Leu Ser Lys Asp Thr Tyr Asp Asp Asp Leu Asp Asn Leu Leu Ala Gln
420 425 430
Ile Gly Asp Gln Tyr Ala Asp Leu Phe Leu Ala Ala Lys Asn Leu Ser
435 440 445
Asp Ala Ile Leu Leu Ser Asp Ile Leu Arg Val Asn Thr Glu Ile Thr
450 455 460
Lys Ala Pro Leu Ser Ala Ser Met Ile Lys Arg Tyr Asp Glu His His
465 470 475 480
Gln Asp Leu Thr Leu Leu Lys Ala Leu Val Arg Gln Gln Leu Pro Glu
485 490 495
Lys Tyr Lys Glu Ile Phe Phe Asp Gln Ser Lys Asn Gly Tyr Ala Gly
500 505 510
Tyr Ile Asp Gly Gly Ala Ser Gln Glu Glu Phe Tyr Lys Phe Ile Lys
515 520 525
Pro Ile Leu Glu Lys Met Asp Gly Thr Glu Glu Leu Leu Val Lys Leu
530 535 540
Asn Arg Glu Asp Leu Leu Arg Lys Gln Arg Thr Phe Asp Asn Gly Ser
545 550 555 560
Ile Pro His Gln Ile His Leu Gly Glu Leu His Ala Ile Leu Arg Arg
565 570 575
Gln Glu Asp Phe Tyr Pro Phe Leu Lys Asp Asn Arg Glu Lys Ile Glu
580 585 590
Lys Ile Leu Thr Phe Arg Ile Pro Tyr Tyr Val Gly Pro Leu Ala Arg
595 600 605
Gly Asn Ser Arg Phe Ala Trp Met Thr Arg Lys Ser Glu Glu Thr Ile
610 615 620
Thr Pro Trp Asn Phe Glu Glu Val Val Asp Lys Gly Ala Ser Ala Gln
625 630 635 640
Ser Phe Ile Glu Arg Met Thr Asn Phe Asp Lys Asn Leu Pro Asn Glu
645 650 655
Lys Val Leu Pro Lys His Ser Leu Leu Tyr Glu Tyr Phe Thr Val Tyr
660 665 670
Asn Glu Leu Thr Lys Val Lys Tyr Val Thr Glu Gly Met Arg Lys Pro
675 680 685
Ala Phe Leu Ser Gly Glu Gln Lys Lys Ala Ile Val Asp Leu Leu Phe
690 695 700
Lys Thr Asn Arg Lys Val Thr Val Lys Gln Leu Lys Glu Asp Tyr Phe
705 710 715 720
Lys Lys Ile Glu Cys Phe Asp Ser Val Glu Ile Ser Gly Val Glu Asp
725 730 735
Arg Phe Asn Ala Ser Leu Gly Thr Tyr His Asp Leu Leu Lys Ile Ile
740 745 750
Lys Asp Lys Asp Phe Leu Asp Asn Glu Glu Asn Glu Asp Ile Leu Glu
755 760 765
Asp Ile Val Leu Thr Leu Thr Leu Phe Glu Asp Arg Glu Met Ile Glu
770 775 780
Glu Arg Leu Lys Thr Tyr Ala His Leu Phe Asp Asp Lys Val Met Lys
785 790 795 800
Gln Leu Lys Arg Arg Arg Tyr Thr Gly Trp Gly Arg Leu Ser Arg Lys
805 810 815
Leu Ile Asn Gly Ile Arg Asp Lys Gln Ser Gly Lys Thr Ile Leu Asp
820 825 830
Phe Leu Lys Ser Asp Gly Phe Ala Asn Arg Asn Phe Met Gln Leu Ile
835 840 845
His Asp Asp Ser Leu Thr Phe Lys Glu Asp Ile Gln Lys Ala Gln Val
850 855 860
Ser Gly Gln Gly Asp Ser Leu His Glu His Ile Ala Asn Leu Ala Gly
865 870 875 880
Ser Pro Ala Ile Lys Lys Gly Ile Leu Gln Thr Val Lys Val Val Asp
885 890 895
Glu Leu Val Lys Val Met Gly Arg His Lys Pro Glu Asn Ile Val Ile
900 905 910
Glu Met Ala Arg Glu Asn Gln Thr Thr Gln Lys Gly Gln Lys Asn Ser
915 920 925
Arg Glu Arg Met Lys Arg Ile Glu Glu Gly Ile Lys Glu Leu Gly Ser
930 935 940
Gln Ile Leu Lys Glu His Pro Val Glu Asn Thr Gln Leu Gln Asn Glu
945 950 955 960
Lys Leu Tyr Leu Tyr Tyr Leu Gln Asn Gly Arg Asp Met Tyr Val Asp
965 970 975
Gln Glu Leu Asp Ile Asn Arg Leu Ser Asp Tyr Asp Val Asp Ala Ile
980 985 990
Val Pro Gln Ser Phe Leu Lys Asp Asp Ser Ile Asp Asn Lys Val Leu
995 1000 1005
Thr Arg Ser Asp Lys Asn Arg Gly Lys Ser Asp Asn Val Pro Ser
1010 1015 1020
Glu Glu Val Val Lys Lys Met Lys Asn Tyr Trp Arg Gln Leu Leu
1025 1030 1035
Asn Ala Lys Leu Ile Thr Gln Arg Lys Phe Asp Asn Leu Thr Lys
1040 1045 1050
Ala Glu Arg Gly Gly Leu Ser Glu Leu Asp Lys Ala Gly Phe Ile
1055 1060 1065
Lys Arg Gln Leu Val Glu Thr Arg Gln Ile Thr Lys His Val Ala
1070 1075 1080
Gln Ile Leu Asp Ser Arg Met Asn Thr Lys Tyr Asp Glu Asn Asp
1085 1090 1095
Lys Leu Ile Arg Glu Val Lys Val Ile Thr Leu Lys Ser Lys Leu
1100 1105 1110
Val Ser Asp Phe Arg Lys Asp Phe Gln Phe Tyr Lys Val Arg Glu
1115 1120 1125
Ile Asn Asn Tyr His His Ala His Asp Ala Tyr Leu Asn Ala Val
1130 1135 1140
Val Gly Thr Ala Leu Ile Lys Lys Tyr Pro Lys Leu Glu Ser Glu
1145 1150 1155
Phe Val Tyr Gly Asp Tyr Lys Val Tyr Asp Val Arg Lys Met Ile
1160 1165 1170
Ala Lys Ser Glu Gln Glu Ile Gly Lys Ala Thr Ala Lys Tyr Phe
1175 1180 1185
Phe Tyr Ser Asn Ile Met Asn Phe Phe Lys Thr Glu Ile Thr Leu
1190 1195 1200
Ala Asn Gly Glu Ile Arg Lys Arg Pro Leu Ile Glu Thr Asn Gly
1205 1210 1215
Glu Thr Gly Glu Ile Val Trp Asp Lys Gly Arg Asp Phe Ala Thr
1220 1225 1230
Val Arg Lys Val Leu Ser Met Pro Gln Val Asn Ile Val Lys Lys
1235 1240 1245
Thr Glu Val Gln Thr Gly Gly Phe Ser Lys Glu Ser Ile Leu Pro
1250 1255 1260
Lys Arg Asn Ser Asp Lys Leu Ile Ala Arg Lys Lys Asp Trp Asp
1265 1270 1275
Pro Lys Lys Tyr Gly Gly Phe Asp Ser Pro Thr Val Ala Tyr Ser
1280 1285 1290
Val Leu Val Val Ala Lys Val Glu Lys Gly Lys Ser Lys Lys Leu
1295 1300 1305
Lys Ser Val Lys Glu Leu Leu Gly Ile Thr Ile Met Glu Arg Ser
1310 1315 1320
Ser Phe Glu Lys Asn Pro Ile Asp Phe Leu Glu Ala Lys Gly Tyr
1325 1330 1335
Lys Glu Val Lys Lys Asp Leu Ile Ile Lys Leu Pro Lys Tyr Ser
1340 1345 1350
Leu Phe Glu Leu Glu Asn Gly Arg Lys Arg Met Leu Ala Ser Ala
1355 1360 1365
Gly Glu Leu Gln Lys Gly Asn Glu Leu Ala Leu Pro Ser Lys Tyr
1370 1375 1380
Val Asn Phe Leu Tyr Leu Ala Ser His Tyr Glu Lys Leu Lys Gly
1385 1390 1395
Ser Pro Glu Asp Asn Glu Gln Lys Gln Leu Phe Val Glu Gln His
1400 1405 1410
Lys His Tyr Leu Asp Glu Ile Ile Glu Gln Ile Ser Glu Phe Ser
1415 1420 1425
Lys Arg Val Ile Leu Ala Asp Ala Asn Leu Asp Lys Val Leu Ser
1430 1435 1440
Ala Tyr Asn Lys His Arg Asp Lys Pro Ile Arg Glu Gln Ala Glu
1445 1450 1455
Asn Ile Ile His Leu Phe Thr Leu Thr Asn Leu Gly Ala Pro Ala
1460 1465 1470
Ala Phe Lys Tyr Phe Asp Thr Thr Ile Asp Arg Lys Arg Tyr Thr
1475 1480 1485
Ser Thr Lys Glu Val Leu Asp Ala Thr Leu Ile His Gln Ser Ile
1490 1495 1500
Thr Gly Leu Tyr Glu Thr Arg Ile Asp Leu Ser Gln Leu Gly Gly
1505 1510 1515
Asp Ser Arg Ala Asp Tyr Pro Tyr Asp Val Pro Asp Tyr Ala Ser
1520 1525 1530
Gly Ser Pro Lys Lys Lys Arg Lys Val Glu Ala Ser Gly Ser Gly
1535 1540 1545
Arg Ala Ser Pro Gly Ile Pro Gly Ser Thr Arg Asn His Asp Gln
1550 1555 1560
Glu Phe Asp Pro Pro Lys Val Tyr Pro Pro Val Pro Ala Glu Lys
1565 1570 1575
Arg Lys Pro Ile Arg Val Leu Ser Leu Phe Asp Gly Ile Ala Thr
1580 1585 1590
Gly Leu Leu Val Leu Lys Asp Leu Gly Ile Gln Val Asp Arg Tyr
1595 1600 1605
Ile Ala Ser Glu Val Cys Glu Asp Ser Ile Thr Val Gly Met Val
1610 1615 1620
Arg His Gln Gly Lys Ile Met Tyr Val Gly Asp Val Arg Ser Val
1625 1630 1635
Thr Gln Lys His Ile Gln Glu Trp Gly Pro Phe Asp Leu Val Ile
1640 1645 1650
Gly Gly Ser Pro Cys Asn Asp Leu Ser Ile Val Asn Pro Ala Arg
1655 1660 1665
Lys Gly Leu Tyr Glu Gly Thr Gly Arg Leu Phe Phe Glu Phe Tyr
1670 1675 1680
Arg Leu Leu His Asp Ala Arg Pro Lys Glu Gly Asp Asp Arg Pro
1685 1690 1695
Phe Phe Trp Leu Phe Glu Asn Val Val Ala Met Gly Val Ser Asp
1700 1705 1710
Lys Arg Asp Ile Ser Arg Phe Leu Glu Ser Asn Pro Val Met Ile
1715 1720 1725
Asp Ala Lys Glu Val Ser Ala Ala His Arg Ala Arg Tyr Phe Trp
1730 1735 1740
Gly Asn Leu Pro Gly Met Asn Arg Pro Leu Ala Ser Thr Val Asn
1745 1750 1755
Asp Lys Leu Glu Leu Gln Glu Cys Leu Glu His Gly Arg Ile Ala
1760 1765 1770
Lys Phe Ser Lys Val Arg Thr Ile Thr Thr Arg Ser Asn Ser Ile
1775 1780 1785
Lys Gln Gly Lys Asp Gln His Phe Pro Val Phe Met Asn Glu Lys
1790 1795 1800
Glu Asp Ile Leu Trp Cys Thr Glu Met Glu Arg Val Phe Gly Phe
1805 1810 1815
Pro Val His Tyr Thr Asp Val Ser Asn Met Ser Arg Leu Ala Arg
1820 1825 1830
Gln Arg Leu Leu Gly Arg Ser Trp Ser Val Pro Val Ile Arg His
1835 1840 1845
Leu Phe Ala Pro Leu Lys Glu Tyr Phe Ala Cys Val Ser Ser Gly
1850 1855 1860
Asn Ser Asn Ala Asn Ser Arg Gly Pro Ser Phe Ser Ser Gly Leu
1865 1870 1875
Val Pro Leu Ser Leu Arg Gly Ser His Met Gly Pro Met Glu Ile
1880 1885 1890
Tyr Lys Thr Val Ser Ala Trp Lys Arg Gln Pro Val Arg Val Leu
1895 1900 1905
Ser Leu Phe Arg Asn Ile Asp Lys Val Leu Lys Ser Leu Gly Phe
1910 1915 1920
Leu Glu Ser Gly Ser Gly Ser Gly Gly Gly Thr Leu Lys Tyr Val
1925 1930 1935
Glu Asp Val Thr Asn Val Val Arg Arg Asp Val Glu Lys Trp Gly
1940 1945 1950
Pro Phe Asp Leu Val Tyr Gly Ser Thr Gln Pro Leu Gly Ser Ser
1955 1960 1965
Cys Asp Arg Cys Pro Gly Trp Tyr Met Phe Gln Phe His Arg Ile
1970 1975 1980
Leu Gln Tyr Ala Leu Pro Arg Gln Glu Ser Gln Arg Pro Phe Phe
1985 1990 1995
Trp Ile Phe Met Asp Asn Leu Leu Leu Thr Glu Asp Asp Gln Glu
2000 2005 2010
Thr Thr Thr Arg Phe Leu Gln Thr Glu Ala Val Thr Leu Gln Asp
2015 2020 2025
Val Arg Gly Arg Asp Tyr Gln Asn Ala Met Arg Val Trp Ser Asn
2030 2035 2040
Ile Pro Gly Leu Lys Ser Lys His Ala Pro Leu Thr Pro Lys Glu
2045 2050 2055
Glu Glu Tyr Leu Gln Ala Gln Val Arg Ser Arg Ser Lys Leu Asp
2060 2065 2070
Ala Pro Lys Val Asp Leu Leu Val Lys Asn Cys Leu Leu Pro Leu
2075 2080 2085
Arg Glu Tyr Phe Lys Tyr Phe Ser Gln Asn Ser Leu Pro Leu Ser
2090 2095 2100
Arg Ala Asp Pro Lys Lys Lys Arg Lys Val Gly Ser Gly Ala Thr
2105 2110 2115
Asn Phe Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro
2120 2125 2130
Gly Pro Ser Glu Leu Ile Lys Glu Asn Met His Met Lys Leu Tyr
2135 2140 2145
Met Glu Gly Thr Val Asp Asn His His Phe Lys Cys Thr Ser Glu
2150 2155 2160
Gly Glu Gly Lys Pro Tyr Glu Gly Thr Gln Thr Met Arg Ile Lys
2165 2170 2175
Val Val Glu Gly Gly Pro Leu Pro Phe Ala Phe Asp Ile Leu Ala
2180 2185 2190
Thr Ser Phe Leu Tyr Gly Ser Lys Thr Phe Ile Asn His Thr Gln
2195 2200 2205
Gly Ile Pro Asp Phe Phe Lys Gln Ser Phe Pro Glu Gly Phe Thr
2210 2215 2220
Trp Glu Arg Val Thr Thr Tyr Glu Asp Gly Gly Val Leu Thr Ala
2225 2230 2235
Thr Gln Asp Thr Ser Leu Gln Asp Gly Cys Leu Ile Tyr Asn Val
2240 2245 2250
Lys Ile Arg Gly Val Asn Phe Thr Ser Asn Gly Pro Val Met Gln
2255 2260 2265
Lys Lys Thr Leu Gly Trp Glu Ala Phe Thr Glu Thr Leu Tyr Pro
2270 2275 2280
Ala Asp Gly Gly Leu Glu Gly Arg Asn Asp Met Ala Leu Lys Leu
2285 2290 2295
Val Gly Gly Ser His Leu Ile Ala Asn Ile Lys Thr Thr Tyr Arg
2300 2305 2310
Ser Lys Lys Pro Ala Lys Asn Leu Lys Met Pro Gly Val Tyr Tyr
2315 2320 2325
Val Asp Tyr Arg Leu Glu Arg Ile Lys Glu Ala Asn Asn Glu Thr
2330 2335 2340
Tyr Val Glu Gln His Glu Val Ala Val Ala Arg Tyr Cys Asp Leu
2345 2350 2355
Pro Ser Lys Leu Gly His Lys Leu Asn
2360 2365
<210> 10
<211> 2389
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетический полипептид
<400> 10
Asp Ala Lys Ser Leu Thr Ala Trp Ser Arg Thr Leu Val Thr Phe Lys
1. 5 10 15
Asp Val Phe Val Asp Phe Thr Arg Glu Glu Trp Lys Leu Leu Asp Thr
20 25 30
Ala Gln Gln Ile Val Tyr Arg Asn Val Met Leu Glu Asn Tyr Lys Asn
35 40 45
Leu Val Ser Leu Gly Tyr Gln Leu Thr Lys Pro Asp Val Ile Leu Arg
50 55 60
Leu Glu Lys Gly Glu Glu Pro Gly Gly Pro Ser Ser Gly Ala Pro Pro
65 70 75 80
Pro Ser Gly Gly Ser Pro Ala Gly Ser Pro Thr Ser Thr Glu Glu Gly
85 90 95
Thr Ser Glu Ser Ala Thr Pro Glu Ser Gly Pro Gly Thr Ser Thr Glu
100 105 110
Pro Ser Glu Gly Ser Ala Pro Gly Ser Pro Ala Gly Ser Pro Thr Ser
115 120 125
Thr Glu Glu Gly Thr Ser Thr Glu Pro Ser Glu Gly Ser Ala Pro Gly
130 135 140
Thr Ser Thr Glu Pro Ser Glu Met Asp Lys Lys Tyr Ser Ile Gly Leu
145 150 155 160
Ala Ile Gly Thr Asn Ser Val Gly Trp Ala Val Ile Thr Asp Glu Tyr
165 170 175
Lys Val Pro Ser Lys Lys Phe Lys Val Leu Gly Asn Thr Asp Arg His
180 185 190
Ser Ile Lys Lys Asn Leu Ile Gly Ala Leu Leu Phe Asp Ser Gly Glu
195 200 205
Thr Ala Glu Ala Thr Arg Leu Lys Arg Thr Ala Arg Arg Arg Tyr Thr
210 215 220
Arg Arg Lys Asn Arg Ile Cys Tyr Leu Gln Glu Ile Phe Ser Asn Glu
225 230 235 240
Met Ala Lys Val Asp Asp Ser Phe Phe His Arg Leu Glu Glu Ser Phe
245 250 255
Leu Val Glu Glu Asp Lys Lys His Glu Arg His Pro Ile Phe Gly Asn
260 265 270
Ile Val Asp Glu Val Ala Tyr His Glu Lys Tyr Pro Thr Ile Tyr His
275 280 285
Leu Arg Lys Lys Leu Val Asp Ser Thr Asp Lys Ala Asp Leu Arg Leu
290 295 300
Ile Tyr Leu Ala Leu Ala His Met Ile Lys Phe Arg Gly His Phe Leu
305 310 315 320
Ile Glu Gly Asp Leu Asn Pro Asp Asn Ser Asp Val Asp Lys Leu Phe
325 330 335
Ile Gln Leu Val Gln Thr Tyr Asn Gln Leu Phe Glu Glu Asn Pro Ile
340 345 350
Asn Ala Ser Gly Val Asp Ala Lys Ala Ile Leu Ser Ala Arg Leu Ser
355 360 365
Lys Ser Arg Arg Leu Glu Asn Leu Ile Ala Gln Leu Pro Gly Glu Lys
370 375 380
Lys Asn Gly Leu Phe Gly Asn Leu Ile Ala Leu Ser Leu Gly Leu Thr
385 390 395 400
Pro Asn Phe Lys Ser Asn Phe Asp Leu Ala Glu Asp Ala Lys Leu Gln
405 410 415
Leu Ser Lys Asp Thr Tyr Asp Asp Asp Leu Asp Asn Leu Leu Ala Gln
420 425 430
Ile Gly Asp Gln Tyr Ala Asp Leu Phe Leu Ala Ala Lys Asn Leu Ser
435 440 445
Asp Ala Ile Leu Leu Ser Asp Ile Leu Arg Val Asn Thr Glu Ile Thr
450 455 460
Lys Ala Pro Leu Ser Ala Ser Met Ile Lys Arg Tyr Asp Glu His His
465 470 475 480
Gln Asp Leu Thr Leu Leu Lys Ala Leu Val Arg Gln Gln Leu Pro Glu
485 490 495
Lys Tyr Lys Glu Ile Phe Phe Asp Gln Ser Lys Asn Gly Tyr Ala Gly
500 505 510
Tyr Ile Asp Gly Gly Ala Ser Gln Glu Glu Phe Tyr Lys Phe Ile Lys
515 520 525
Pro Ile Leu Glu Lys Met Asp Gly Thr Glu Glu Leu Leu Val Lys Leu
530 535 540
Asn Arg Glu Asp Leu Leu Arg Lys Gln Arg Thr Phe Asp Asn Gly Ser
545 550 555 560
Ile Pro His Gln Ile His Leu Gly Glu Leu His Ala Ile Leu Arg Arg
565 570 575
Gln Glu Asp Phe Tyr Pro Phe Leu Lys Asp Asn Arg Glu Lys Ile Glu
580 585 590
Lys Ile Leu Thr Phe Arg Ile Pro Tyr Tyr Val Gly Pro Leu Ala Arg
595 600 605
Gly Asn Ser Arg Phe Ala Trp Met Thr Arg Lys Ser Glu Glu Thr Ile
610 615 620
Thr Pro Trp Asn Phe Glu Glu Val Val Asp Lys Gly Ala Ser Ala Gln
625 630 635 640
Ser Phe Ile Glu Arg Met Thr Asn Phe Asp Lys Asn Leu Pro Asn Glu
645 650 655
Lys Val Leu Pro Lys His Ser Leu Leu Tyr Glu Tyr Phe Thr Val Tyr
660 665 670
Asn Glu Leu Thr Lys Val Lys Tyr Val Thr Glu Gly Met Arg Lys Pro
675 680 685
Ala Phe Leu Ser Gly Glu Gln Lys Lys Ala Ile Val Asp Leu Leu Phe
690 695 700
Lys Thr Asn Arg Lys Val Thr Val Lys Gln Leu Lys Glu Asp Tyr Phe
705 710 715 720
Lys Lys Ile Glu Cys Phe Asp Ser Val Glu Ile Ser Gly Val Glu Asp
725 730 735
Arg Phe Asn Ala Ser Leu Gly Thr Tyr His Asp Leu Leu Lys Ile Ile
740 745 750
Lys Asp Lys Asp Phe Leu Asp Asn Glu Glu Asn Glu Asp Ile Leu Glu
755 760 765
Asp Ile Val Leu Thr Leu Thr Leu Phe Glu Asp Arg Glu Met Ile Glu
770 775 780
Glu Arg Leu Lys Thr Tyr Ala His Leu Phe Asp Asp Lys Val Met Lys
785 790 795 800
Gln Leu Lys Arg Arg Arg Tyr Thr Gly Trp Gly Arg Leu Ser Arg Lys
805 810 815
Leu Ile Asn Gly Ile Arg Asp Lys Gln Ser Gly Lys Thr Ile Leu Asp
820 825 830
Phe Leu Lys Ser Asp Gly Phe Ala Asn Arg Asn Phe Met Gln Leu Ile
835 840 845
His Asp Asp Ser Leu Thr Phe Lys Glu Asp Ile Gln Lys Ala Gln Val
850 855 860
Ser Gly Gln Gly Asp Ser Leu His Glu His Ile Ala Asn Leu Ala Gly
865 870 875 880
Ser Pro Ala Ile Lys Lys Gly Ile Leu Gln Thr Val Lys Val Val Asp
885 890 895
Glu Leu Val Lys Val Met Gly Arg His Lys Pro Glu Asn Ile Val Ile
900 905 910
Glu Met Ala Arg Glu Asn Gln Thr Thr Gln Lys Gly Gln Lys Asn Ser
915 920 925
Arg Glu Arg Met Lys Arg Ile Glu Glu Gly Ile Lys Glu Leu Gly Ser
930 935 940
Gln Ile Leu Lys Glu His Pro Val Glu Asn Thr Gln Leu Gln Asn Glu
945 950 955 960
Lys Leu Tyr Leu Tyr Tyr Leu Gln Asn Gly Arg Asp Met Tyr Val Asp
965 970 975
Gln Glu Leu Asp Ile Asn Arg Leu Ser Asp Tyr Asp Val Asp Ala Ile
980 985 990
Val Pro Gln Ser Phe Leu Lys Asp Asp Ser Ile Asp Asn Lys Val Leu
995 1000 1005
Thr Arg Ser Asp Lys Asn Arg Gly Lys Ser Asp Asn Val Pro Ser
1010 1015 1020
Glu Glu Val Val Lys Lys Met Lys Asn Tyr Trp Arg Gln Leu Leu
1025 1030 1035
Asn Ala Lys Leu Ile Thr Gln Arg Lys Phe Asp Asn Leu Thr Lys
1040 1045 1050
Ala Glu Arg Gly Gly Leu Ser Glu Leu Asp Lys Ala Gly Phe Ile
1055 1060 1065
Lys Arg Gln Leu Val Glu Thr Arg Gln Ile Thr Lys His Val Ala
1070 1075 1080
Gln Ile Leu Asp Ser Arg Met Asn Thr Lys Tyr Asp Glu Asn Asp
1085 1090 1095
Lys Leu Ile Arg Glu Val Lys Val Ile Thr Leu Lys Ser Lys Leu
1100 1105 1110
Val Ser Asp Phe Arg Lys Asp Phe Gln Phe Tyr Lys Val Arg Glu
1115 1120 1125
Ile Asn Asn Tyr His His Ala His Asp Ala Tyr Leu Asn Ala Val
1130 1135 1140
Val Gly Thr Ala Leu Ile Lys Lys Tyr Pro Lys Leu Glu Ser Glu
1145 1150 1155
Phe Val Tyr Gly Asp Tyr Lys Val Tyr Asp Val Arg Lys Met Ile
1160 1165 1170
Ala Lys Ser Glu Gln Glu Ile Gly Lys Ala Thr Ala Lys Tyr Phe
1175 1180 1185
Phe Tyr Ser Asn Ile Met Asn Phe Phe Lys Thr Glu Ile Thr Leu
1190 1195 1200
Ala Asn Gly Glu Ile Arg Lys Arg Pro Leu Ile Glu Thr Asn Gly
1205 1210 1215
Glu Thr Gly Glu Ile Val Trp Asp Lys Gly Arg Asp Phe Ala Thr
1220 1225 1230
Val Arg Lys Val Leu Ser Met Pro Gln Val Asn Ile Val Lys Lys
1235 1240 1245
Thr Glu Val Gln Thr Gly Gly Phe Ser Lys Glu Ser Ile Leu Pro
1250 1255 1260
Lys Arg Asn Ser Asp Lys Leu Ile Ala Arg Lys Lys Asp Trp Asp
1265 1270 1275
Pro Lys Lys Tyr Gly Gly Phe Asp Ser Pro Thr Val Ala Tyr Ser
1280 1285 1290
Val Leu Val Val Ala Lys Val Glu Lys Gly Lys Ser Lys Lys Leu
1295 1300 1305
Lys Ser Val Lys Glu Leu Leu Gly Ile Thr Ile Met Glu Arg Ser
1310 1315 1320
Ser Phe Glu Lys Asn Pro Ile Asp Phe Leu Glu Ala Lys Gly Tyr
1325 1330 1335
Lys Glu Val Lys Lys Asp Leu Ile Ile Lys Leu Pro Lys Tyr Ser
1340 1345 1350
Leu Phe Glu Leu Glu Asn Gly Arg Lys Arg Met Leu Ala Ser Ala
1355 1360 1365
Gly Glu Leu Gln Lys Gly Asn Glu Leu Ala Leu Pro Ser Lys Tyr
1370 1375 1380
Val Asn Phe Leu Tyr Leu Ala Ser His Tyr Glu Lys Leu Lys Gly
1385 1390 1395
Ser Pro Glu Asp Asn Glu Gln Lys Gln Leu Phe Val Glu Gln His
1400 1405 1410
Lys His Tyr Leu Asp Glu Ile Ile Glu Gln Ile Ser Glu Phe Ser
1415 1420 1425
Lys Arg Val Ile Leu Ala Asp Ala Asn Leu Asp Lys Val Leu Ser
1430 1435 1440
Ala Tyr Asn Lys His Arg Asp Lys Pro Ile Arg Glu Gln Ala Glu
1445 1450 1455
Asn Ile Ile His Leu Phe Thr Leu Thr Asn Leu Gly Ala Pro Ala
1460 1465 1470
Ala Phe Lys Tyr Phe Asp Thr Thr Ile Asp Arg Lys Arg Tyr Thr
1475 1480 1485
Ser Thr Lys Glu Val Leu Asp Ala Thr Leu Ile His Gln Ser Ile
1490 1495 1500
Thr Gly Leu Tyr Glu Thr Arg Ile Asp Leu Ser Gln Leu Gly Gly
1505 1510 1515
Asp Ser Arg Ala Asp Tyr Pro Tyr Asp Val Pro Asp Tyr Ala Ser
1520 1525 1530
Gly Ser Pro Lys Lys Lys Arg Lys Val Glu Ala Ser Gly Ser Gly
1535 1540 1545
Arg Ala Ser Pro Gly Ile Pro Gly Ser Thr Arg Asn His Asp Gln
1550 1555 1560
Glu Phe Asp Pro Pro Lys Val Tyr Pro Pro Val Pro Ala Glu Lys
1565 1570 1575
Arg Lys Pro Ile Arg Val Leu Ser Leu Phe Asp Gly Ile Ala Thr
1580 1585 1590
Gly Leu Leu Val Leu Lys Asp Leu Gly Ile Gln Val Asp Arg Tyr
1595 1600 1605
Ile Ala Ser Glu Val Cys Glu Asp Ser Ile Thr Val Gly Met Val
1610 1615 1620
Arg His Gln Gly Lys Ile Met Tyr Val Gly Asp Val Arg Ser Val
1625 1630 1635
Thr Gln Lys His Ile Gln Glu Trp Gly Pro Phe Asp Leu Val Ile
1640 1645 1650
Gly Gly Ser Pro Cys Asn Asp Leu Ser Ile Val Asn Pro Ala Arg
1655 1660 1665
Lys Gly Leu Tyr Glu Gly Thr Gly Arg Leu Phe Phe Glu Phe Tyr
1670 1675 1680
Arg Leu Leu His Asp Ala Arg Pro Lys Glu Gly Asp Asp Arg Pro
1685 1690 1695
Phe Phe Trp Leu Phe Glu Asn Val Val Ala Met Gly Val Ser Asp
1700 1705 1710
Lys Arg Asp Ile Ser Arg Phe Leu Glu Ser Asn Pro Val Met Ile
1715 1720 1725
Asp Ala Lys Glu Val Ser Ala Ala His Arg Ala Arg Tyr Phe Trp
1730 1735 1740
Gly Asn Leu Pro Gly Met Asn Arg Pro Leu Ala Ser Thr Val Asn
1745 1750 1755
Asp Lys Leu Glu Leu Gln Glu Cys Leu Glu His Gly Arg Ile Ala
1760 1765 1770
Lys Phe Ser Lys Val Arg Thr Ile Thr Thr Arg Ser Asn Ser Ile
1775 1780 1785
Lys Gln Gly Lys Asp Gln His Phe Pro Val Phe Met Asn Glu Lys
1790 1795 1800
Glu Asp Ile Leu Trp Cys Thr Glu Met Glu Arg Val Phe Gly Phe
1805 1810 1815
Pro Val His Tyr Thr Asp Val Ser Asn Met Ser Arg Leu Ala Arg
1820 1825 1830
Gln Arg Leu Leu Gly Arg Ser Trp Ser Val Pro Val Ile Arg His
1835 1840 1845
Leu Phe Ala Pro Leu Lys Glu Tyr Phe Ala Cys Val Ser Ser Gly
1850 1855 1860
Asn Ser Asn Ala Asn Ser Arg Gly Pro Ser Phe Ser Ser Gly Leu
1865 1870 1875
Val Pro Leu Ser Leu Arg Gly Ser His Met Gly Pro Met Glu Ile
1880 1885 1890
Tyr Lys Thr Val Ser Ala Trp Lys Arg Gln Pro Val Arg Val Leu
1895 1900 1905
Ser Leu Phe Arg Asn Ile Asp Lys Val Leu Lys Ser Leu Gly Phe
1910 1915 1920
Leu Glu Ser Gly Ser Gly Ser Gly Gly Gly Thr Leu Lys Tyr Val
1925 1930 1935
Glu Asp Val Thr Asn Val Val Arg Arg Asp Val Glu Lys Trp Gly
1940 1945 1950
Pro Phe Asp Leu Val Tyr Gly Ser Thr Gln Pro Leu Gly Ser Ser
1955 1960 1965
Cys Asp Arg Cys Pro Gly Trp Tyr Met Phe Gln Phe His Arg Ile
1970 1975 1980
Leu Gln Tyr Ala Leu Pro Arg Gln Glu Ser Gln Arg Pro Phe Phe
1985 1990 1995
Trp Ile Phe Met Asp Asn Leu Leu Leu Thr Glu Asp Asp Gln Glu
2000 2005 2010
Thr Thr Thr Arg Phe Leu Gln Thr Glu Ala Val Thr Leu Gln Asp
2015 2020 2025
Val Arg Gly Arg Asp Tyr Gln Asn Ala Met Arg Val Trp Ser Asn
2030 2035 2040
Ile Pro Gly Leu Lys Ser Lys His Ala Pro Leu Thr Pro Lys Glu
2045 2050 2055
Glu Glu Tyr Leu Gln Ala Gln Val Arg Ser Arg Ser Lys Leu Asp
2060 2065 2070
Ala Pro Lys Val Asp Leu Leu Val Lys Asn Cys Leu Leu Pro Leu
2075 2080 2085
Arg Glu Tyr Phe Lys Tyr Phe Ser Gln Asn Ser Leu Pro Leu Ser
2090 2095 2100
Arg Ala Asp Pro Lys Lys Lys Arg Lys Val Gly Ser Gly Ala Thr
2105 2110 2115
Asn Phe Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro
2120 2125 2130
Gly Pro Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu Lys Gln Ala
2135 2140 2145
Gly Asp Val Glu Glu Asn Pro Gly Pro Ser Glu Leu Ile Lys Glu
2150 2155 2160
Asn Met His Met Lys Leu Tyr Met Glu Gly Thr Val Asp Asn His
2165 2170 2175
His Phe Lys Cys Thr Ser Glu Gly Glu Gly Lys Pro Tyr Glu Gly
2180 2185 2190
Thr Gln Thr Met Arg Ile Lys Val Val Glu Gly Gly Pro Leu Pro
2195 2200 2205
Phe Ala Phe Asp Ile Leu Ala Thr Ser Phe Leu Tyr Gly Ser Lys
2210 2215 2220
Thr Phe Ile Asn His Thr Gln Gly Ile Pro Asp Phe Phe Lys Gln
2225 2230 2235
Ser Phe Pro Glu Gly Phe Thr Trp Glu Arg Val Thr Thr Tyr Glu
2240 2245 2250
Asp Gly Gly Val Leu Thr Ala Thr Gln Asp Thr Ser Leu Gln Asp
2255 2260 2265
Gly Cys Leu Ile Tyr Asn Val Lys Ile Arg Gly Val Asn Phe Thr
2270 2275 2280
Ser Asn Gly Pro Val Met Gln Lys Lys Thr Leu Gly Trp Glu Ala
2285 2290 2295
Phe Thr Glu Thr Leu Tyr Pro Ala Asp Gly Gly Leu Glu Gly Arg
2300 2305 2310
Asn Asp Met Ala Leu Lys Leu Val Gly Gly Ser His Leu Ile Ala
2315 2320 2325
Asn Ile Lys Thr Thr Tyr Arg Ser Lys Lys Pro Ala Lys Asn Leu
2330 2335 2340
Lys Met Pro Gly Val Tyr Tyr Val Asp Tyr Arg Leu Glu Arg Ile
2345 2350 2355
Lys Glu Ala Asn Asn Glu Thr Tyr Val Glu Gln His Glu Val Ala
2360 2365 2370
Val Ala Arg Tyr Cys Asp Leu Pro Ser Lys Leu Gly His Lys Leu
2375 2380 2385
Asn
<210> 11
<211> 2369
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетический полипептид
<400> 11
Asp Ala Lys Ser Leu Thr Ala Trp Ser Arg Thr Leu Val Thr Phe Lys
1. 5 10 15
Asp Val Phe Val Asp Phe Thr Arg Glu Glu Trp Lys Leu Leu Asp Thr
20 25 30
Ala Gln Gln Ile Val Tyr Arg Asn Val Met Leu Glu Asn Tyr Lys Asn
35 40 45
Leu Val Ser Leu Gly Tyr Gln Leu Thr Lys Pro Asp Val Ile Leu Arg
50 55 60
Leu Glu Lys Gly Glu Glu Pro Ser Gly Ser Glu Thr Pro Gly Thr Ser
65 70 75 80
Glu Ser Ala Thr Pro Glu Ser Met Asp Lys Lys Tyr Ser Ile Gly Leu
85 90 95
Ala Ile Gly Thr Asn Ser Val Gly Trp Ala Val Ile Thr Asp Glu Tyr
100 105 110
Lys Val Pro Ser Lys Lys Phe Lys Val Leu Gly Asn Thr Asp Arg His
115 120 125
Ser Ile Lys Lys Asn Leu Ile Gly Ala Leu Leu Phe Asp Ser Gly Glu
130 135 140
Thr Ala Glu Ala Thr Arg Leu Lys Arg Thr Ala Arg Arg Arg Tyr Thr
145 150 155 160
Arg Arg Lys Asn Arg Ile Cys Tyr Leu Gln Glu Ile Phe Ser Asn Glu
165 170 175
Met Ala Lys Val Asp Asp Ser Phe Phe His Arg Leu Glu Glu Ser Phe
180 185 190
Leu Val Glu Glu Asp Lys Lys His Glu Arg His Pro Ile Phe Gly Asn
195 200 205
Ile Val Asp Glu Val Ala Tyr His Glu Lys Tyr Pro Thr Ile Tyr His
210 215 220
Leu Arg Lys Lys Leu Val Asp Ser Thr Asp Lys Ala Asp Leu Arg Leu
225 230 235 240
Ile Tyr Leu Ala Leu Ala His Met Ile Lys Phe Arg Gly His Phe Leu
245 250 255
Ile Glu Gly Asp Leu Asn Pro Asp Asn Ser Asp Val Asp Lys Leu Phe
260 265 270
Ile Gln Leu Val Gln Thr Tyr Asn Gln Leu Phe Glu Glu Asn Pro Ile
275 280 285
Asn Ala Ser Gly Val Asp Ala Lys Ala Ile Leu Ser Ala Arg Leu Ser
290 295 300
Lys Ser Arg Arg Leu Glu Asn Leu Ile Ala Gln Leu Pro Gly Glu Lys
305 310 315 320
Lys Asn Gly Leu Phe Gly Asn Leu Ile Ala Leu Ser Leu Gly Leu Thr
325 330 335
Pro Asn Phe Lys Ser Asn Phe Asp Leu Ala Glu Asp Ala Lys Leu Gln
340 345 350
Leu Ser Lys Asp Thr Tyr Asp Asp Asp Leu Asp Asn Leu Leu Ala Gln
355 360 365
Ile Gly Asp Gln Tyr Ala Asp Leu Phe Leu Ala Ala Lys Asn Leu Ser
370 375 380
Asp Ala Ile Leu Leu Ser Asp Ile Leu Arg Val Asn Thr Glu Ile Thr
385 390 395 400
Lys Ala Pro Leu Ser Ala Ser Met Ile Lys Arg Tyr Asp Glu His His
405 410 415
Gln Asp Leu Thr Leu Leu Lys Ala Leu Val Arg Gln Gln Leu Pro Glu
420 425 430
Lys Tyr Lys Glu Ile Phe Phe Asp Gln Ser Lys Asn Gly Tyr Ala Gly
435 440 445
Tyr Ile Asp Gly Gly Ala Ser Gln Glu Glu Phe Tyr Lys Phe Ile Lys
450 455 460
Pro Ile Leu Glu Lys Met Asp Gly Thr Glu Glu Leu Leu Val Lys Leu
465 470 475 480
Asn Arg Glu Asp Leu Leu Arg Lys Gln Arg Thr Phe Asp Asn Gly Ser
485 490 495
Ile Pro His Gln Ile His Leu Gly Glu Leu His Ala Ile Leu Arg Arg
500 505 510
Gln Glu Asp Phe Tyr Pro Phe Leu Lys Asp Asn Arg Glu Lys Ile Glu
515 520 525
Lys Ile Leu Thr Phe Arg Ile Pro Tyr Tyr Val Gly Pro Leu Ala Arg
530 535 540
Gly Asn Ser Arg Phe Ala Trp Met Thr Arg Lys Ser Glu Glu Thr Ile
545 550 555 560
Thr Pro Trp Asn Phe Glu Glu Val Val Asp Lys Gly Ala Ser Ala Gln
565 570 575
Ser Phe Ile Glu Arg Met Thr Asn Phe Asp Lys Asn Leu Pro Asn Glu
580 585 590
Lys Val Leu Pro Lys His Ser Leu Leu Tyr Glu Tyr Phe Thr Val Tyr
595 600 605
Asn Glu Leu Thr Lys Val Lys Tyr Val Thr Glu Gly Met Arg Lys Pro
610 615 620
Ala Phe Leu Ser Gly Glu Gln Lys Lys Ala Ile Val Asp Leu Leu Phe
625 630 635 640
Lys Thr Asn Arg Lys Val Thr Val Lys Gln Leu Lys Glu Asp Tyr Phe
645 650 655
Lys Lys Ile Glu Cys Phe Asp Ser Val Glu Ile Ser Gly Val Glu Asp
660 665 670
Arg Phe Asn Ala Ser Leu Gly Thr Tyr His Asp Leu Leu Lys Ile Ile
675 680 685
Lys Asp Lys Asp Phe Leu Asp Asn Glu Glu Asn Glu Asp Ile Leu Glu
690 695 700
Asp Ile Val Leu Thr Leu Thr Leu Phe Glu Asp Arg Glu Met Ile Glu
705 710 715 720
Glu Arg Leu Lys Thr Tyr Ala His Leu Phe Asp Asp Lys Val Met Lys
725 730 735
Gln Leu Lys Arg Arg Arg Tyr Thr Gly Trp Gly Arg Leu Ser Arg Lys
740 745 750
Leu Ile Asn Gly Ile Arg Asp Lys Gln Ser Gly Lys Thr Ile Leu Asp
755 760 765
Phe Leu Lys Ser Asp Gly Phe Ala Asn Arg Asn Phe Met Gln Leu Ile
770 775 780
His Asp Asp Ser Leu Thr Phe Lys Glu Asp Ile Gln Lys Ala Gln Val
785 790 795 800
Ser Gly Gln Gly Asp Ser Leu His Glu His Ile Ala Asn Leu Ala Gly
805 810 815
Ser Pro Ala Ile Lys Lys Gly Ile Leu Gln Thr Val Lys Val Val Asp
820 825 830
Glu Leu Val Lys Val Met Gly Arg His Lys Pro Glu Asn Ile Val Ile
835 840 845
Glu Met Ala Arg Glu Asn Gln Thr Thr Gln Lys Gly Gln Lys Asn Ser
850 855 860
Arg Glu Arg Met Lys Arg Ile Glu Glu Gly Ile Lys Glu Leu Gly Ser
865 870 875 880
Gln Ile Leu Lys Glu His Pro Val Glu Asn Thr Gln Leu Gln Asn Glu
885 890 895
Lys Leu Tyr Leu Tyr Tyr Leu Gln Asn Gly Arg Asp Met Tyr Val Asp
900 905 910
Gln Glu Leu Asp Ile Asn Arg Leu Ser Asp Tyr Asp Val Asp Ala Ile
915 920 925
Val Pro Gln Ser Phe Leu Lys Asp Asp Ser Ile Asp Asn Lys Val Leu
930 935 940
Thr Arg Ser Asp Lys Asn Arg Gly Lys Ser Asp Asn Val Pro Ser Glu
945 950 955 960
Glu Val Val Lys Lys Met Lys Asn Tyr Trp Arg Gln Leu Leu Asn Ala
965 970 975
Lys Leu Ile Thr Gln Arg Lys Phe Asp Asn Leu Thr Lys Ala Glu Arg
980 985 990
Gly Gly Leu Ser Glu Leu Asp Lys Ala Gly Phe Ile Lys Arg Gln Leu
995 1000 1005
Val Glu Thr Arg Gln Ile Thr Lys His Val Ala Gln Ile Leu Asp
1010 1015 1020
Ser Arg Met Asn Thr Lys Tyr Asp Glu Asn Asp Lys Leu Ile Arg
1025 1030 1035
Glu Val Lys Val Ile Thr Leu Lys Ser Lys Leu Val Ser Asp Phe
1040 1045 1050
Arg Lys Asp Phe Gln Phe Tyr Lys Val Arg Glu Ile Asn Asn Tyr
1055 1060 1065
His His Ala His Asp Ala Tyr Leu Asn Ala Val Val Gly Thr Ala
1070 1075 1080
Leu Ile Lys Lys Tyr Pro Lys Leu Glu Ser Glu Phe Val Tyr Gly
1085 1090 1095
Asp Tyr Lys Val Tyr Asp Val Arg Lys Met Ile Ala Lys Ser Glu
1100 1105 1110
Gln Glu Ile Gly Lys Ala Thr Ala Lys Tyr Phe Phe Tyr Ser Asn
1115 1120 1125
Ile Met Asn Phe Phe Lys Thr Glu Ile Thr Leu Ala Asn Gly Glu
1130 1135 1140
Ile Arg Lys Arg Pro Leu Ile Glu Thr Asn Gly Glu Thr Gly Glu
1145 1150 1155
Ile Val Trp Asp Lys Gly Arg Asp Phe Ala Thr Val Arg Lys Val
1160 1165 1170
Leu Ser Met Pro Gln Val Asn Ile Val Lys Lys Thr Glu Val Gln
1175 1180 1185
Thr Gly Gly Phe Ser Lys Glu Ser Ile Leu Pro Lys Arg Asn Ser
1190 1195 1200
Asp Lys Leu Ile Ala Arg Lys Lys Asp Trp Asp Pro Lys Lys Tyr
1205 1210 1215
Gly Gly Phe Asp Ser Pro Thr Val Ala Tyr Ser Val Leu Val Val
1220 1225 1230
Ala Lys Val Glu Lys Gly Lys Ser Lys Lys Leu Lys Ser Val Lys
1235 1240 1245
Glu Leu Leu Gly Ile Thr Ile Met Glu Arg Ser Ser Phe Glu Lys
1250 1255 1260
Asn Pro Ile Asp Phe Leu Glu Ala Lys Gly Tyr Lys Glu Val Lys
1265 1270 1275
Lys Asp Leu Ile Ile Lys Leu Pro Lys Tyr Ser Leu Phe Glu Leu
1280 1285 1290
Glu Asn Gly Arg Lys Arg Met Leu Ala Ser Ala Gly Glu Leu Gln
1295 1300 1305
Lys Gly Asn Glu Leu Ala Leu Pro Ser Lys Tyr Val Asn Phe Leu
1310 1315 1320
Tyr Leu Ala Ser His Tyr Glu Lys Leu Lys Gly Ser Pro Glu Asp
1325 1330 1335
Asn Glu Gln Lys Gln Leu Phe Val Glu Gln His Lys His Tyr Leu
1340 1345 1350
Asp Glu Ile Ile Glu Gln Ile Ser Glu Phe Ser Lys Arg Val Ile
1355 1360 1365
Leu Ala Asp Ala Asn Leu Asp Lys Val Leu Ser Ala Tyr Asn Lys
1370 1375 1380
His Arg Asp Lys Pro Ile Arg Glu Gln Ala Glu Asn Ile Ile His
1385 1390 1395
Leu Phe Thr Leu Thr Asn Leu Gly Ala Pro Ala Ala Phe Lys Tyr
1400 1405 1410
Phe Asp Thr Thr Ile Asp Arg Lys Arg Tyr Thr Ser Thr Lys Glu
1415 1420 1425
Val Leu Asp Ala Thr Leu Ile His Gln Ser Ile Thr Gly Leu Tyr
1430 1435 1440
Glu Thr Arg Ile Asp Leu Ser Gln Leu Gly Gly Asp Ser Arg Ala
1445 1450 1455
Asp Tyr Pro Tyr Asp Val Pro Asp Tyr Ala Ser Gly Ser Pro Lys
1460 1465 1470
Lys Lys Arg Lys Val Ser Pro Gly Gly Gly Pro Ser Ser Gly Ala
1475 1480 1485
Pro Pro Pro Ser Gly Gly Ser Pro Ala Gly Ser Pro Thr Ser Thr
1490 1495 1500
Glu Glu Gly Thr Ser Glu Ser Ala Thr Pro Glu Ser Gly Pro Gly
1505 1510 1515
Thr Ser Thr Glu Pro Ser Glu Gly Ser Ala Pro Gly Ser Pro Ala
1520 1525 1530
Gly Ser Pro Thr Ser Thr Glu Glu Gly Thr Ser Thr Glu Pro Ser
1535 1540 1545
Glu Gly Ser Ala Pro Gly Thr Ser Thr Glu Pro Ser Glu Asn His
1550 1555 1560
Asp Gln Glu Phe Asp Pro Pro Lys Val Tyr Pro Pro Val Pro Ala
1565 1570 1575
Glu Lys Arg Lys Pro Ile Arg Val Leu Ser Leu Phe Asp Gly Ile
1580 1585 1590
Ala Thr Gly Leu Leu Val Leu Lys Asp Leu Gly Ile Gln Val Asp
1595 1600 1605
Arg Tyr Ile Ala Ser Glu Val Cys Glu Asp Ser Ile Thr Val Gly
1610 1615 1620
Met Val Arg His Gln Gly Lys Ile Met Tyr Val Gly Asp Val Arg
1625 1630 1635
Ser Val Thr Gln Lys His Ile Gln Glu Trp Gly Pro Phe Asp Leu
1640 1645 1650
Val Ile Gly Gly Ser Pro Cys Asn Asp Leu Ser Ile Val Asn Pro
1655 1660 1665
Ala Arg Lys Gly Leu Tyr Glu Gly Thr Gly Arg Leu Phe Phe Glu
1670 1675 1680
Phe Tyr Arg Leu Leu His Asp Ala Arg Pro Lys Glu Gly Asp Asp
1685 1690 1695
Arg Pro Phe Phe Trp Leu Phe Glu Asn Val Val Ala Met Gly Val
1700 1705 1710
Ser Asp Lys Arg Asp Ile Ser Arg Phe Leu Glu Ser Asn Pro Val
1715 1720 1725
Met Ile Asp Ala Lys Glu Val Ser Ala Ala His Arg Ala Arg Tyr
1730 1735 1740
Phe Trp Gly Asn Leu Pro Gly Met Asn Arg Pro Leu Ala Ser Thr
1745 1750 1755
Val Asn Asp Lys Leu Glu Leu Gln Glu Cys Leu Glu His Gly Arg
1760 1765 1770
Ile Ala Lys Phe Ser Lys Val Arg Thr Ile Thr Thr Arg Ser Asn
1775 1780 1785
Ser Ile Lys Gln Gly Lys Asp Gln His Phe Pro Val Phe Met Asn
1790 1795 1800
Glu Lys Glu Asp Ile Leu Trp Cys Thr Glu Met Glu Arg Val Phe
1805 1810 1815
Gly Phe Pro Val His Tyr Thr Asp Val Ser Asn Met Ser Arg Leu
1820 1825 1830
Ala Arg Gln Arg Leu Leu Gly Arg Ser Trp Ser Val Pro Val Ile
1835 1840 1845
Arg His Leu Phe Ala Pro Leu Lys Glu Tyr Phe Ala Cys Val Ser
1850 1855 1860
Ser Gly Asn Ser Asn Ala Asn Ser Arg Gly Pro Ser Phe Ser Ser
1865 1870 1875
Gly Leu Val Pro Leu Ser Leu Arg Gly Ser His Met Gly Pro Met
1880 1885 1890
Glu Ile Tyr Lys Thr Val Ser Ala Trp Lys Arg Gln Pro Val Arg
1895 1900 1905
Val Leu Ser Leu Phe Arg Asn Ile Asp Lys Val Leu Lys Ser Leu
1910 1915 1920
Gly Phe Leu Glu Ser Gly Ser Gly Ser Gly Gly Gly Thr Leu Lys
1925 1930 1935
Tyr Val Glu Asp Val Thr Asn Val Val Arg Arg Asp Val Glu Lys
1940 1945 1950
Trp Gly Pro Phe Asp Leu Val Tyr Gly Ser Thr Gln Pro Leu Gly
1955 1960 1965
Ser Ser Cys Asp Arg Cys Pro Gly Trp Tyr Met Phe Gln Phe His
1970 1975 1980
Arg Ile Leu Gln Tyr Ala Leu Pro Arg Gln Glu Ser Gln Arg Pro
1985 1990 1995
Phe Phe Trp Ile Phe Met Asp Asn Leu Leu Leu Thr Glu Asp Asp
2000 2005 2010
Gln Glu Thr Thr Thr Arg Phe Leu Gln Thr Glu Ala Val Thr Leu
2015 2020 2025
Gln Asp Val Arg Gly Arg Asp Tyr Gln Asn Ala Met Arg Val Trp
2030 2035 2040
Ser Asn Ile Pro Gly Leu Lys Ser Lys His Ala Pro Leu Thr Pro
2045 2050 2055
Lys Glu Glu Glu Tyr Leu Gln Ala Gln Val Arg Ser Arg Ser Lys
2060 2065 2070
Leu Asp Ala Pro Lys Val Asp Leu Leu Val Lys Asn Cys Leu Leu
2075 2080 2085
Pro Leu Arg Glu Tyr Phe Lys Tyr Phe Ser Gln Asn Ser Leu Pro
2090 2095 2100
Leu Ser Arg Ala Asp Pro Lys Lys Lys Arg Lys Val Gly Ser Gly
2105 2110 2115
Ala Thr Asn Phe Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu
2120 2125 2130
Asn Pro Gly Pro Ser Glu Leu Ile Lys Glu Asn Met His Met Lys
2135 2140 2145
Leu Tyr Met Glu Gly Thr Val Asp Asn His His Phe Lys Cys Thr
2150 2155 2160
Ser Glu Gly Glu Gly Lys Pro Tyr Glu Gly Thr Gln Thr Met Arg
2165 2170 2175
Ile Lys Val Val Glu Gly Gly Pro Leu Pro Phe Ala Phe Asp Ile
2180 2185 2190
Leu Ala Thr Ser Phe Leu Tyr Gly Ser Lys Thr Phe Ile Asn His
2195 2200 2205
Thr Gln Gly Ile Pro Asp Phe Phe Lys Gln Ser Phe Pro Glu Gly
2210 2215 2220
Phe Thr Trp Glu Arg Val Thr Thr Tyr Glu Asp Gly Gly Val Leu
2225 2230 2235
Thr Ala Thr Gln Asp Thr Ser Leu Gln Asp Gly Cys Leu Ile Tyr
2240 2245 2250
Asn Val Lys Ile Arg Gly Val Asn Phe Thr Ser Asn Gly Pro Val
2255 2260 2265
Met Gln Lys Lys Thr Leu Gly Trp Glu Ala Phe Thr Glu Thr Leu
2270 2275 2280
Tyr Pro Ala Asp Gly Gly Leu Glu Gly Arg Asn Asp Met Ala Leu
2285 2290 2295
Lys Leu Val Gly Gly Ser His Leu Ile Ala Asn Ile Lys Thr Thr
2300 2305 2310
Tyr Arg Ser Lys Lys Pro Ala Lys Asn Leu Lys Met Pro Gly Val
2315 2320 2325
Tyr Tyr Val Asp Tyr Arg Leu Glu Arg Ile Lys Glu Ala Asn Asn
2330 2335 2340
Glu Thr Tyr Val Glu Gln His Glu Val Ala Val Ala Arg Tyr Cys
2345 2350 2355
Asp Leu Pro Ser Lys Leu Gly His Lys Leu Asn
2360 2365
<210> 12
<211> 2391
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетический полипептид
<400> 12
Asp Ala Lys Ser Leu Thr Ala Trp Ser Arg Thr Leu Val Thr Phe Lys
1. 5 10 15
Asp Val Phe Val Asp Phe Thr Arg Glu Glu Trp Lys Leu Leu Asp Thr
20 25 30
Ala Gln Gln Ile Val Tyr Arg Asn Val Met Leu Glu Asn Tyr Lys Asn
35 40 45
Leu Val Ser Leu Gly Tyr Gln Leu Thr Lys Pro Asp Val Ile Leu Arg
50 55 60
Leu Glu Lys Gly Glu Glu Pro Ser Gly Ser Glu Thr Pro Gly Thr Ser
65 70 75 80
Glu Ser Ala Thr Pro Glu Ser Met Asp Lys Lys Tyr Ser Ile Gly Leu
85 90 95
Ala Ile Gly Thr Asn Ser Val Gly Trp Ala Val Ile Thr Asp Glu Tyr
100 105 110
Lys Val Pro Ser Lys Lys Phe Lys Val Leu Gly Asn Thr Asp Arg His
115 120 125
Ser Ile Lys Lys Asn Leu Ile Gly Ala Leu Leu Phe Asp Ser Gly Glu
130 135 140
Thr Ala Glu Ala Thr Arg Leu Lys Arg Thr Ala Arg Arg Arg Tyr Thr
145 150 155 160
Arg Arg Lys Asn Arg Ile Cys Tyr Leu Gln Glu Ile Phe Ser Asn Glu
165 170 175
Met Ala Lys Val Asp Asp Ser Phe Phe His Arg Leu Glu Glu Ser Phe
180 185 190
Leu Val Glu Glu Asp Lys Lys His Glu Arg His Pro Ile Phe Gly Asn
195 200 205
Ile Val Asp Glu Val Ala Tyr His Glu Lys Tyr Pro Thr Ile Tyr His
210 215 220
Leu Arg Lys Lys Leu Val Asp Ser Thr Asp Lys Ala Asp Leu Arg Leu
225 230 235 240
Ile Tyr Leu Ala Leu Ala His Met Ile Lys Phe Arg Gly His Phe Leu
245 250 255
Ile Glu Gly Asp Leu Asn Pro Asp Asn Ser Asp Val Asp Lys Leu Phe
260 265 270
Ile Gln Leu Val Gln Thr Tyr Asn Gln Leu Phe Glu Glu Asn Pro Ile
275 280 285
Asn Ala Ser Gly Val Asp Ala Lys Ala Ile Leu Ser Ala Arg Leu Ser
290 295 300
Lys Ser Arg Arg Leu Glu Asn Leu Ile Ala Gln Leu Pro Gly Glu Lys
305 310 315 320
Lys Asn Gly Leu Phe Gly Asn Leu Ile Ala Leu Ser Leu Gly Leu Thr
325 330 335
Pro Asn Phe Lys Ser Asn Phe Asp Leu Ala Glu Asp Ala Lys Leu Gln
340 345 350
Leu Ser Lys Asp Thr Tyr Asp Asp Asp Leu Asp Asn Leu Leu Ala Gln
355 360 365
Ile Gly Asp Gln Tyr Ala Asp Leu Phe Leu Ala Ala Lys Asn Leu Ser
370 375 380
Asp Ala Ile Leu Leu Ser Asp Ile Leu Arg Val Asn Thr Glu Ile Thr
385 390 395 400
Lys Ala Pro Leu Ser Ala Ser Met Ile Lys Arg Tyr Asp Glu His His
405 410 415
Gln Asp Leu Thr Leu Leu Lys Ala Leu Val Arg Gln Gln Leu Pro Glu
420 425 430
Lys Tyr Lys Glu Ile Phe Phe Asp Gln Ser Lys Asn Gly Tyr Ala Gly
435 440 445
Tyr Ile Asp Gly Gly Ala Ser Gln Glu Glu Phe Tyr Lys Phe Ile Lys
450 455 460
Pro Ile Leu Glu Lys Met Asp Gly Thr Glu Glu Leu Leu Val Lys Leu
465 470 475 480
Asn Arg Glu Asp Leu Leu Arg Lys Gln Arg Thr Phe Asp Asn Gly Ser
485 490 495
Ile Pro His Gln Ile His Leu Gly Glu Leu His Ala Ile Leu Arg Arg
500 505 510
Gln Glu Asp Phe Tyr Pro Phe Leu Lys Asp Asn Arg Glu Lys Ile Glu
515 520 525
Lys Ile Leu Thr Phe Arg Ile Pro Tyr Tyr Val Gly Pro Leu Ala Arg
530 535 540
Gly Asn Ser Arg Phe Ala Trp Met Thr Arg Lys Ser Glu Glu Thr Ile
545 550 555 560
Thr Pro Trp Asn Phe Glu Glu Val Val Asp Lys Gly Ala Ser Ala Gln
565 570 575
Ser Phe Ile Glu Arg Met Thr Asn Phe Asp Lys Asn Leu Pro Asn Glu
580 585 590
Lys Val Leu Pro Lys His Ser Leu Leu Tyr Glu Tyr Phe Thr Val Tyr
595 600 605
Asn Glu Leu Thr Lys Val Lys Tyr Val Thr Glu Gly Met Arg Lys Pro
610 615 620
Ala Phe Leu Ser Gly Glu Gln Lys Lys Ala Ile Val Asp Leu Leu Phe
625 630 635 640
Lys Thr Asn Arg Lys Val Thr Val Lys Gln Leu Lys Glu Asp Tyr Phe
645 650 655
Lys Lys Ile Glu Cys Phe Asp Ser Val Glu Ile Ser Gly Val Glu Asp
660 665 670
Arg Phe Asn Ala Ser Leu Gly Thr Tyr His Asp Leu Leu Lys Ile Ile
675 680 685
Lys Asp Lys Asp Phe Leu Asp Asn Glu Glu Asn Glu Asp Ile Leu Glu
690 695 700
Asp Ile Val Leu Thr Leu Thr Leu Phe Glu Asp Arg Glu Met Ile Glu
705 710 715 720
Glu Arg Leu Lys Thr Tyr Ala His Leu Phe Asp Asp Lys Val Met Lys
725 730 735
Gln Leu Lys Arg Arg Arg Tyr Thr Gly Trp Gly Arg Leu Ser Arg Lys
740 745 750
Leu Ile Asn Gly Ile Arg Asp Lys Gln Ser Gly Lys Thr Ile Leu Asp
755 760 765
Phe Leu Lys Ser Asp Gly Phe Ala Asn Arg Asn Phe Met Gln Leu Ile
770 775 780
His Asp Asp Ser Leu Thr Phe Lys Glu Asp Ile Gln Lys Ala Gln Val
785 790 795 800
Ser Gly Gln Gly Asp Ser Leu His Glu His Ile Ala Asn Leu Ala Gly
805 810 815
Ser Pro Ala Ile Lys Lys Gly Ile Leu Gln Thr Val Lys Val Val Asp
820 825 830
Glu Leu Val Lys Val Met Gly Arg His Lys Pro Glu Asn Ile Val Ile
835 840 845
Glu Met Ala Arg Glu Asn Gln Thr Thr Gln Lys Gly Gln Lys Asn Ser
850 855 860
Arg Glu Arg Met Lys Arg Ile Glu Glu Gly Ile Lys Glu Leu Gly Ser
865 870 875 880
Gln Ile Leu Lys Glu His Pro Val Glu Asn Thr Gln Leu Gln Asn Glu
885 890 895
Lys Leu Tyr Leu Tyr Tyr Leu Gln Asn Gly Arg Asp Met Tyr Val Asp
900 905 910
Gln Glu Leu Asp Ile Asn Arg Leu Ser Asp Tyr Asp Val Asp Ala Ile
915 920 925
Val Pro Gln Ser Phe Leu Lys Asp Asp Ser Ile Asp Asn Lys Val Leu
930 935 940
Thr Arg Ser Asp Lys Asn Arg Gly Lys Ser Asp Asn Val Pro Ser Glu
945 950 955 960
Glu Val Val Lys Lys Met Lys Asn Tyr Trp Arg Gln Leu Leu Asn Ala
965 970 975
Lys Leu Ile Thr Gln Arg Lys Phe Asp Asn Leu Thr Lys Ala Glu Arg
980 985 990
Gly Gly Leu Ser Glu Leu Asp Lys Ala Gly Phe Ile Lys Arg Gln Leu
995 1000 1005
Val Glu Thr Arg Gln Ile Thr Lys His Val Ala Gln Ile Leu Asp
1010 1015 1020
Ser Arg Met Asn Thr Lys Tyr Asp Glu Asn Asp Lys Leu Ile Arg
1025 1030 1035
Glu Val Lys Val Ile Thr Leu Lys Ser Lys Leu Val Ser Asp Phe
1040 1045 1050
Arg Lys Asp Phe Gln Phe Tyr Lys Val Arg Glu Ile Asn Asn Tyr
1055 1060 1065
His His Ala His Asp Ala Tyr Leu Asn Ala Val Val Gly Thr Ala
1070 1075 1080
Leu Ile Lys Lys Tyr Pro Lys Leu Glu Ser Glu Phe Val Tyr Gly
1085 1090 1095
Asp Tyr Lys Val Tyr Asp Val Arg Lys Met Ile Ala Lys Ser Glu
1100 1105 1110
Gln Glu Ile Gly Lys Ala Thr Ala Lys Tyr Phe Phe Tyr Ser Asn
1115 1120 1125
Ile Met Asn Phe Phe Lys Thr Glu Ile Thr Leu Ala Asn Gly Glu
1130 1135 1140
Ile Arg Lys Arg Pro Leu Ile Glu Thr Asn Gly Glu Thr Gly Glu
1145 1150 1155
Ile Val Trp Asp Lys Gly Arg Asp Phe Ala Thr Val Arg Lys Val
1160 1165 1170
Leu Ser Met Pro Gln Val Asn Ile Val Lys Lys Thr Glu Val Gln
1175 1180 1185
Thr Gly Gly Phe Ser Lys Glu Ser Ile Leu Pro Lys Arg Asn Ser
1190 1195 1200
Asp Lys Leu Ile Ala Arg Lys Lys Asp Trp Asp Pro Lys Lys Tyr
1205 1210 1215
Gly Gly Phe Asp Ser Pro Thr Val Ala Tyr Ser Val Leu Val Val
1220 1225 1230
Ala Lys Val Glu Lys Gly Lys Ser Lys Lys Leu Lys Ser Val Lys
1235 1240 1245
Glu Leu Leu Gly Ile Thr Ile Met Glu Arg Ser Ser Phe Glu Lys
1250 1255 1260
Asn Pro Ile Asp Phe Leu Glu Ala Lys Gly Tyr Lys Glu Val Lys
1265 1270 1275
Lys Asp Leu Ile Ile Lys Leu Pro Lys Tyr Ser Leu Phe Glu Leu
1280 1285 1290
Glu Asn Gly Arg Lys Arg Met Leu Ala Ser Ala Gly Glu Leu Gln
1295 1300 1305
Lys Gly Asn Glu Leu Ala Leu Pro Ser Lys Tyr Val Asn Phe Leu
1310 1315 1320
Tyr Leu Ala Ser His Tyr Glu Lys Leu Lys Gly Ser Pro Glu Asp
1325 1330 1335
Asn Glu Gln Lys Gln Leu Phe Val Glu Gln His Lys His Tyr Leu
1340 1345 1350
Asp Glu Ile Ile Glu Gln Ile Ser Glu Phe Ser Lys Arg Val Ile
1355 1360 1365
Leu Ala Asp Ala Asn Leu Asp Lys Val Leu Ser Ala Tyr Asn Lys
1370 1375 1380
His Arg Asp Lys Pro Ile Arg Glu Gln Ala Glu Asn Ile Ile His
1385 1390 1395
Leu Phe Thr Leu Thr Asn Leu Gly Ala Pro Ala Ala Phe Lys Tyr
1400 1405 1410
Phe Asp Thr Thr Ile Asp Arg Lys Arg Tyr Thr Ser Thr Lys Glu
1415 1420 1425
Val Leu Asp Ala Thr Leu Ile His Gln Ser Ile Thr Gly Leu Tyr
1430 1435 1440
Glu Thr Arg Ile Asp Leu Ser Gln Leu Gly Gly Asp Ser Arg Ala
1445 1450 1455
Asp Tyr Pro Tyr Asp Val Pro Asp Tyr Ala Ser Gly Ser Pro Lys
1460 1465 1470
Lys Lys Arg Lys Val Ser Pro Gly Gly Gly Pro Ser Ser Gly Ala
1475 1480 1485
Pro Pro Pro Ser Gly Gly Ser Pro Ala Gly Ser Pro Thr Ser Thr
1490 1495 1500
Glu Glu Gly Thr Ser Glu Ser Ala Thr Pro Glu Ser Gly Pro Gly
1505 1510 1515
Thr Ser Thr Glu Pro Ser Glu Gly Ser Ala Pro Gly Ser Pro Ala
1520 1525 1530
Gly Ser Pro Thr Ser Thr Glu Glu Gly Thr Ser Thr Glu Pro Ser
1535 1540 1545
Glu Gly Ser Ala Pro Gly Thr Ser Thr Glu Pro Ser Glu Asn His
1550 1555 1560
Asp Gln Glu Phe Asp Pro Pro Lys Val Tyr Pro Pro Val Pro Ala
1565 1570 1575
Glu Lys Arg Lys Pro Ile Arg Val Leu Ser Leu Phe Asp Gly Ile
1580 1585 1590
Ala Thr Gly Leu Leu Val Leu Lys Asp Leu Gly Ile Gln Val Asp
1595 1600 1605
Arg Tyr Ile Ala Ser Glu Val Cys Glu Asp Ser Ile Thr Val Gly
1610 1615 1620
Met Val Arg His Gln Gly Lys Ile Met Tyr Val Gly Asp Val Arg
1625 1630 1635
Ser Val Thr Gln Lys His Ile Gln Glu Trp Gly Pro Phe Asp Leu
1640 1645 1650
Val Ile Gly Gly Ser Pro Cys Asn Asp Leu Ser Ile Val Asn Pro
1655 1660 1665
Ala Arg Lys Gly Leu Tyr Glu Gly Thr Gly Arg Leu Phe Phe Glu
1670 1675 1680
Phe Tyr Arg Leu Leu His Asp Ala Arg Pro Lys Glu Gly Asp Asp
1685 1690 1695
Arg Pro Phe Phe Trp Leu Phe Glu Asn Val Val Ala Met Gly Val
1700 1705 1710
Ser Asp Lys Arg Asp Ile Ser Arg Phe Leu Glu Ser Asn Pro Val
1715 1720 1725
Met Ile Asp Ala Lys Glu Val Ser Ala Ala His Arg Ala Arg Tyr
1730 1735 1740
Phe Trp Gly Asn Leu Pro Gly Met Asn Arg Pro Leu Ala Ser Thr
1745 1750 1755
Val Asn Asp Lys Leu Glu Leu Gln Glu Cys Leu Glu His Gly Arg
1760 1765 1770
Ile Ala Lys Phe Ser Lys Val Arg Thr Ile Thr Thr Arg Ser Asn
1775 1780 1785
Ser Ile Lys Gln Gly Lys Asp Gln His Phe Pro Val Phe Met Asn
1790 1795 1800
Glu Lys Glu Asp Ile Leu Trp Cys Thr Glu Met Glu Arg Val Phe
1805 1810 1815
Gly Phe Pro Val His Tyr Thr Asp Val Ser Asn Met Ser Arg Leu
1820 1825 1830
Ala Arg Gln Arg Leu Leu Gly Arg Ser Trp Ser Val Pro Val Ile
1835 1840 1845
Arg His Leu Phe Ala Pro Leu Lys Glu Tyr Phe Ala Cys Val Ser
1850 1855 1860
Ser Gly Asn Ser Asn Ala Asn Ser Arg Gly Pro Ser Phe Ser Ser
1865 1870 1875
Gly Leu Val Pro Leu Ser Leu Arg Gly Ser His Met Gly Pro Met
1880 1885 1890
Glu Ile Tyr Lys Thr Val Ser Ala Trp Lys Arg Gln Pro Val Arg
1895 1900 1905
Val Leu Ser Leu Phe Arg Asn Ile Asp Lys Val Leu Lys Ser Leu
1910 1915 1920
Gly Phe Leu Glu Ser Gly Ser Gly Ser Gly Gly Gly Thr Leu Lys
1925 1930 1935
Tyr Val Glu Asp Val Thr Asn Val Val Arg Arg Asp Val Glu Lys
1940 1945 1950
Trp Gly Pro Phe Asp Leu Val Tyr Gly Ser Thr Gln Pro Leu Gly
1955 1960 1965
Ser Ser Cys Asp Arg Cys Pro Gly Trp Tyr Met Phe Gln Phe His
1970 1975 1980
Arg Ile Leu Gln Tyr Ala Leu Pro Arg Gln Glu Ser Gln Arg Pro
1985 1990 1995
Phe Phe Trp Ile Phe Met Asp Asn Leu Leu Leu Thr Glu Asp Asp
2000 2005 2010
Gln Glu Thr Thr Thr Arg Phe Leu Gln Thr Glu Ala Val Thr Leu
2015 2020 2025
Gln Asp Val Arg Gly Arg Asp Tyr Gln Asn Ala Met Arg Val Trp
2030 2035 2040
Ser Asn Ile Pro Gly Leu Lys Ser Lys His Ala Pro Leu Thr Pro
2045 2050 2055
Lys Glu Glu Glu Tyr Leu Gln Ala Gln Val Arg Ser Arg Ser Lys
2060 2065 2070
Leu Asp Ala Pro Lys Val Asp Leu Leu Val Lys Asn Cys Leu Leu
2075 2080 2085
Pro Leu Arg Glu Tyr Phe Lys Tyr Phe Ser Gln Asn Ser Leu Pro
2090 2095 2100
Leu Ser Arg Ala Asp Pro Lys Lys Lys Arg Lys Val Gly Ser Gly
2105 2110 2115
Ala Thr Asn Phe Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu
2120 2125 2130
Asn Pro Gly Pro Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu Lys
2135 2140 2145
Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Ser Glu Leu Ile
2150 2155 2160
Lys Glu Asn Met His Met Lys Leu Tyr Met Glu Gly Thr Val Asp
2165 2170 2175
Asn His His Phe Lys Cys Thr Ser Glu Gly Glu Gly Lys Pro Tyr
2180 2185 2190
Glu Gly Thr Gln Thr Met Arg Ile Lys Val Val Glu Gly Gly Pro
2195 2200 2205
Leu Pro Phe Ala Phe Asp Ile Leu Ala Thr Ser Phe Leu Tyr Gly
2210 2215 2220
Ser Lys Thr Phe Ile Asn His Thr Gln Gly Ile Pro Asp Phe Phe
2225 2230 2235
Lys Gln Ser Phe Pro Glu Gly Phe Thr Trp Glu Arg Val Thr Thr
2240 2245 2250
Tyr Glu Asp Gly Gly Val Leu Thr Ala Thr Gln Asp Thr Ser Leu
2255 2260 2265
Gln Asp Gly Cys Leu Ile Tyr Asn Val Lys Ile Arg Gly Val Asn
2270 2275 2280
Phe Thr Ser Asn Gly Pro Val Met Gln Lys Lys Thr Leu Gly Trp
2285 2290 2295
Glu Ala Phe Thr Glu Thr Leu Tyr Pro Ala Asp Gly Gly Leu Glu
2300 2305 2310
Gly Arg Asn Asp Met Ala Leu Lys Leu Val Gly Gly Ser His Leu
2315 2320 2325
Ile Ala Asn Ile Lys Thr Thr Tyr Arg Ser Lys Lys Pro Ala Lys
2330 2335 2340
Asn Leu Lys Met Pro Gly Val Tyr Tyr Val Asp Tyr Arg Leu Glu
2345 2350 2355
Arg Ile Lys Glu Ala Asn Asn Glu Thr Tyr Val Glu Gln His Glu
2360 2365 2370
Val Ala Val Ala Arg Tyr Cys Asp Leu Pro Ser Lys Leu Gly His
2375 2380 2385
Lys Leu Asn
2390
<210> 13
<211> 2369
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетический полипептид
<400> 13
Asp Ala Lys Ser Leu Thr Ala Trp Ser Arg Thr Leu Val Thr Phe Lys
1. 5 10 15
Asp Val Phe Val Asp Phe Thr Arg Glu Glu Trp Lys Leu Leu Asp Thr
20 25 30
Ala Gln Gln Ile Val Tyr Arg Asn Val Met Leu Glu Asn Tyr Lys Asn
35 40 45
Leu Val Ser Leu Gly Tyr Gln Leu Thr Lys Pro Asp Val Ile Leu Arg
50 55 60
Leu Glu Lys Gly Glu Glu Pro Gly Gly Pro Ser Ser Gly Ala Pro Pro
65 70 75 80
Pro Ser Gly Gly Ser Pro Ala Gly Ser Pro Thr Ser Thr Glu Glu Gly
85 90 95
Thr Ser Glu Ser Ala Thr Pro Glu Ser Gly Pro Gly Thr Ser Thr Glu
100 105 110
Pro Ser Glu Gly Ser Ala Pro Gly Ser Pro Ala Gly Ser Pro Thr Ser
115 120 125
Thr Glu Glu Gly Thr Ser Thr Glu Pro Ser Glu Gly Ser Ala Pro Gly
130 135 140
Thr Ser Thr Glu Pro Ser Glu Met Asp Lys Lys Tyr Ser Ile Gly Leu
145 150 155 160
Ala Ile Gly Thr Asn Ser Val Gly Trp Ala Val Ile Thr Asp Glu Tyr
165 170 175
Lys Val Pro Ser Lys Lys Phe Lys Val Leu Gly Asn Thr Asp Arg His
180 185 190
Ser Ile Lys Lys Asn Leu Ile Gly Ala Leu Leu Phe Asp Ser Gly Glu
195 200 205
Thr Ala Glu Ala Thr Arg Leu Lys Arg Thr Ala Arg Arg Arg Tyr Thr
210 215 220
Arg Arg Lys Asn Arg Ile Cys Tyr Leu Gln Glu Ile Phe Ser Asn Glu
225 230 235 240
Met Ala Lys Val Asp Asp Ser Phe Phe His Arg Leu Glu Glu Ser Phe
245 250 255
Leu Val Glu Glu Asp Lys Lys His Glu Arg His Pro Ile Phe Gly Asn
260 265 270
Ile Val Asp Glu Val Ala Tyr His Glu Lys Tyr Pro Thr Ile Tyr His
275 280 285
Leu Arg Lys Lys Leu Val Asp Ser Thr Asp Lys Ala Asp Leu Arg Leu
290 295 300
Ile Tyr Leu Ala Leu Ala His Met Ile Lys Phe Arg Gly His Phe Leu
305 310 315 320
Ile Glu Gly Asp Leu Asn Pro Asp Asn Ser Asp Val Asp Lys Leu Phe
325 330 335
Ile Gln Leu Val Gln Thr Tyr Asn Gln Leu Phe Glu Glu Asn Pro Ile
340 345 350
Asn Ala Ser Gly Val Asp Ala Lys Ala Ile Leu Ser Ala Arg Leu Ser
355 360 365
Lys Ser Arg Arg Leu Glu Asn Leu Ile Ala Gln Leu Pro Gly Glu Lys
370 375 380
Lys Asn Gly Leu Phe Gly Asn Leu Ile Ala Leu Ser Leu Gly Leu Thr
385 390 395 400
Pro Asn Phe Lys Ser Asn Phe Asp Leu Ala Glu Asp Ala Lys Leu Gln
405 410 415
Leu Ser Lys Asp Thr Tyr Asp Asp Asp Leu Asp Asn Leu Leu Ala Gln
420 425 430
Ile Gly Asp Gln Tyr Ala Asp Leu Phe Leu Ala Ala Lys Asn Leu Ser
435 440 445
Asp Ala Ile Leu Leu Ser Asp Ile Leu Arg Val Asn Thr Glu Ile Thr
450 455 460
Lys Ala Pro Leu Ser Ala Ser Met Ile Lys Arg Tyr Asp Glu His His
465 470 475 480
Gln Asp Leu Thr Leu Leu Lys Ala Leu Val Arg Gln Gln Leu Pro Glu
485 490 495
Lys Tyr Lys Glu Ile Phe Phe Asp Gln Ser Lys Asn Gly Tyr Ala Gly
500 505 510
Tyr Ile Asp Gly Gly Ala Ser Gln Glu Glu Phe Tyr Lys Phe Ile Lys
515 520 525
Pro Ile Leu Glu Lys Met Asp Gly Thr Glu Glu Leu Leu Val Lys Leu
530 535 540
Asn Arg Glu Asp Leu Leu Arg Lys Gln Arg Thr Phe Asp Asn Gly Ser
545 550 555 560
Ile Pro His Gln Ile His Leu Gly Glu Leu His Ala Ile Leu Arg Arg
565 570 575
Gln Glu Asp Phe Tyr Pro Phe Leu Lys Asp Asn Arg Glu Lys Ile Glu
580 585 590
Lys Ile Leu Thr Phe Arg Ile Pro Tyr Tyr Val Gly Pro Leu Ala Arg
595 600 605
Gly Asn Ser Arg Phe Ala Trp Met Thr Arg Lys Ser Glu Glu Thr Ile
610 615 620
Thr Pro Trp Asn Phe Glu Glu Val Val Asp Lys Gly Ala Ser Ala Gln
625 630 635 640
Ser Phe Ile Glu Arg Met Thr Asn Phe Asp Lys Asn Leu Pro Asn Glu
645 650 655
Lys Val Leu Pro Lys His Ser Leu Leu Tyr Glu Tyr Phe Thr Val Tyr
660 665 670
Asn Glu Leu Thr Lys Val Lys Tyr Val Thr Glu Gly Met Arg Lys Pro
675 680 685
Ala Phe Leu Ser Gly Glu Gln Lys Lys Ala Ile Val Asp Leu Leu Phe
690 695 700
Lys Thr Asn Arg Lys Val Thr Val Lys Gln Leu Lys Glu Asp Tyr Phe
705 710 715 720
Lys Lys Ile Glu Cys Phe Asp Ser Val Glu Ile Ser Gly Val Glu Asp
725 730 735
Arg Phe Asn Ala Ser Leu Gly Thr Tyr His Asp Leu Leu Lys Ile Ile
740 745 750
Lys Asp Lys Asp Phe Leu Asp Asn Glu Glu Asn Glu Asp Ile Leu Glu
755 760 765
Asp Ile Val Leu Thr Leu Thr Leu Phe Glu Asp Arg Glu Met Ile Glu
770 775 780
Glu Arg Leu Lys Thr Tyr Ala His Leu Phe Asp Asp Lys Val Met Lys
785 790 795 800
Gln Leu Lys Arg Arg Arg Tyr Thr Gly Trp Gly Arg Leu Ser Arg Lys
805 810 815
Leu Ile Asn Gly Ile Arg Asp Lys Gln Ser Gly Lys Thr Ile Leu Asp
820 825 830
Phe Leu Lys Ser Asp Gly Phe Ala Asn Arg Asn Phe Met Gln Leu Ile
835 840 845
His Asp Asp Ser Leu Thr Phe Lys Glu Asp Ile Gln Lys Ala Gln Val
850 855 860
Ser Gly Gln Gly Asp Ser Leu His Glu His Ile Ala Asn Leu Ala Gly
865 870 875 880
Ser Pro Ala Ile Lys Lys Gly Ile Leu Gln Thr Val Lys Val Val Asp
885 890 895
Glu Leu Val Lys Val Met Gly Arg His Lys Pro Glu Asn Ile Val Ile
900 905 910
Glu Met Ala Arg Glu Asn Gln Thr Thr Gln Lys Gly Gln Lys Asn Ser
915 920 925
Arg Glu Arg Met Lys Arg Ile Glu Glu Gly Ile Lys Glu Leu Gly Ser
930 935 940
Gln Ile Leu Lys Glu His Pro Val Glu Asn Thr Gln Leu Gln Asn Glu
945 950 955 960
Lys Leu Tyr Leu Tyr Tyr Leu Gln Asn Gly Arg Asp Met Tyr Val Asp
965 970 975
Gln Glu Leu Asp Ile Asn Arg Leu Ser Asp Tyr Asp Val Asp Ala Ile
980 985 990
Val Pro Gln Ser Phe Leu Lys Asp Asp Ser Ile Asp Asn Lys Val Leu
995 1000 1005
Thr Arg Ser Asp Lys Asn Arg Gly Lys Ser Asp Asn Val Pro Ser
1010 1015 1020
Glu Glu Val Val Lys Lys Met Lys Asn Tyr Trp Arg Gln Leu Leu
1025 1030 1035
Asn Ala Lys Leu Ile Thr Gln Arg Lys Phe Asp Asn Leu Thr Lys
1040 1045 1050
Ala Glu Arg Gly Gly Leu Ser Glu Leu Asp Lys Ala Gly Phe Ile
1055 1060 1065
Lys Arg Gln Leu Val Glu Thr Arg Gln Ile Thr Lys His Val Ala
1070 1075 1080
Gln Ile Leu Asp Ser Arg Met Asn Thr Lys Tyr Asp Glu Asn Asp
1085 1090 1095
Lys Leu Ile Arg Glu Val Lys Val Ile Thr Leu Lys Ser Lys Leu
1100 1105 1110
Val Ser Asp Phe Arg Lys Asp Phe Gln Phe Tyr Lys Val Arg Glu
1115 1120 1125
Ile Asn Asn Tyr His His Ala His Asp Ala Tyr Leu Asn Ala Val
1130 1135 1140
Val Gly Thr Ala Leu Ile Lys Lys Tyr Pro Lys Leu Glu Ser Glu
1145 1150 1155
Phe Val Tyr Gly Asp Tyr Lys Val Tyr Asp Val Arg Lys Met Ile
1160 1165 1170
Ala Lys Ser Glu Gln Glu Ile Gly Lys Ala Thr Ala Lys Tyr Phe
1175 1180 1185
Phe Tyr Ser Asn Ile Met Asn Phe Phe Lys Thr Glu Ile Thr Leu
1190 1195 1200
Ala Asn Gly Glu Ile Arg Lys Arg Pro Leu Ile Glu Thr Asn Gly
1205 1210 1215
Glu Thr Gly Glu Ile Val Trp Asp Lys Gly Arg Asp Phe Ala Thr
1220 1225 1230
Val Arg Lys Val Leu Ser Met Pro Gln Val Asn Ile Val Lys Lys
1235 1240 1245
Thr Glu Val Gln Thr Gly Gly Phe Ser Lys Glu Ser Ile Leu Pro
1250 1255 1260
Lys Arg Asn Ser Asp Lys Leu Ile Ala Arg Lys Lys Asp Trp Asp
1265 1270 1275
Pro Lys Lys Tyr Gly Gly Phe Asp Ser Pro Thr Val Ala Tyr Ser
1280 1285 1290
Val Leu Val Val Ala Lys Val Glu Lys Gly Lys Ser Lys Lys Leu
1295 1300 1305
Lys Ser Val Lys Glu Leu Leu Gly Ile Thr Ile Met Glu Arg Ser
1310 1315 1320
Ser Phe Glu Lys Asn Pro Ile Asp Phe Leu Glu Ala Lys Gly Tyr
1325 1330 1335
Lys Glu Val Lys Lys Asp Leu Ile Ile Lys Leu Pro Lys Tyr Ser
1340 1345 1350
Leu Phe Glu Leu Glu Asn Gly Arg Lys Arg Met Leu Ala Ser Ala
1355 1360 1365
Gly Glu Leu Gln Lys Gly Asn Glu Leu Ala Leu Pro Ser Lys Tyr
1370 1375 1380
Val Asn Phe Leu Tyr Leu Ala Ser His Tyr Glu Lys Leu Lys Gly
1385 1390 1395
Ser Pro Glu Asp Asn Glu Gln Lys Gln Leu Phe Val Glu Gln His
1400 1405 1410
Lys His Tyr Leu Asp Glu Ile Ile Glu Gln Ile Ser Glu Phe Ser
1415 1420 1425
Lys Arg Val Ile Leu Ala Asp Ala Asn Leu Asp Lys Val Leu Ser
1430 1435 1440
Ala Tyr Asn Lys His Arg Asp Lys Pro Ile Arg Glu Gln Ala Glu
1445 1450 1455
Asn Ile Ile His Leu Phe Thr Leu Thr Asn Leu Gly Ala Pro Ala
1460 1465 1470
Ala Phe Lys Tyr Phe Asp Thr Thr Ile Asp Arg Lys Arg Tyr Thr
1475 1480 1485
Ser Thr Lys Glu Val Leu Asp Ala Thr Leu Ile His Gln Ser Ile
1490 1495 1500
Thr Gly Leu Tyr Glu Thr Arg Ile Asp Leu Ser Gln Leu Gly Gly
1505 1510 1515
Asp Ser Arg Ala Asp Tyr Pro Tyr Asp Val Pro Asp Tyr Ala Ser
1520 1525 1530
Gly Ser Pro Lys Lys Lys Arg Lys Val Ser Pro Gly Ser Gly Ser
1535 1540 1545
Glu Thr Pro Gly Thr Ser Glu Ser Ala Thr Pro Glu Ser Asn His
1550 1555 1560
Asp Gln Glu Phe Asp Pro Pro Lys Val Tyr Pro Pro Val Pro Ala
1565 1570 1575
Glu Lys Arg Lys Pro Ile Arg Val Leu Ser Leu Phe Asp Gly Ile
1580 1585 1590
Ala Thr Gly Leu Leu Val Leu Lys Asp Leu Gly Ile Gln Val Asp
1595 1600 1605
Arg Tyr Ile Ala Ser Glu Val Cys Glu Asp Ser Ile Thr Val Gly
1610 1615 1620
Met Val Arg His Gln Gly Lys Ile Met Tyr Val Gly Asp Val Arg
1625 1630 1635
Ser Val Thr Gln Lys His Ile Gln Glu Trp Gly Pro Phe Asp Leu
1640 1645 1650
Val Ile Gly Gly Ser Pro Cys Asn Asp Leu Ser Ile Val Asn Pro
1655 1660 1665
Ala Arg Lys Gly Leu Tyr Glu Gly Thr Gly Arg Leu Phe Phe Glu
1670 1675 1680
Phe Tyr Arg Leu Leu His Asp Ala Arg Pro Lys Glu Gly Asp Asp
1685 1690 1695
Arg Pro Phe Phe Trp Leu Phe Glu Asn Val Val Ala Met Gly Val
1700 1705 1710
Ser Asp Lys Arg Asp Ile Ser Arg Phe Leu Glu Ser Asn Pro Val
1715 1720 1725
Met Ile Asp Ala Lys Glu Val Ser Ala Ala His Arg Ala Arg Tyr
1730 1735 1740
Phe Trp Gly Asn Leu Pro Gly Met Asn Arg Pro Leu Ala Ser Thr
1745 1750 1755
Val Asn Asp Lys Leu Glu Leu Gln Glu Cys Leu Glu His Gly Arg
1760 1765 1770
Ile Ala Lys Phe Ser Lys Val Arg Thr Ile Thr Thr Arg Ser Asn
1775 1780 1785
Ser Ile Lys Gln Gly Lys Asp Gln His Phe Pro Val Phe Met Asn
1790 1795 1800
Glu Lys Glu Asp Ile Leu Trp Cys Thr Glu Met Glu Arg Val Phe
1805 1810 1815
Gly Phe Pro Val His Tyr Thr Asp Val Ser Asn Met Ser Arg Leu
1820 1825 1830
Ala Arg Gln Arg Leu Leu Gly Arg Ser Trp Ser Val Pro Val Ile
1835 1840 1845
Arg His Leu Phe Ala Pro Leu Lys Glu Tyr Phe Ala Cys Val Ser
1850 1855 1860
Ser Gly Asn Ser Asn Ala Asn Ser Arg Gly Pro Ser Phe Ser Ser
1865 1870 1875
Gly Leu Val Pro Leu Ser Leu Arg Gly Ser His Met Gly Pro Met
1880 1885 1890
Glu Ile Tyr Lys Thr Val Ser Ala Trp Lys Arg Gln Pro Val Arg
1895 1900 1905
Val Leu Ser Leu Phe Arg Asn Ile Asp Lys Val Leu Lys Ser Leu
1910 1915 1920
Gly Phe Leu Glu Ser Gly Ser Gly Ser Gly Gly Gly Thr Leu Lys
1925 1930 1935
Tyr Val Glu Asp Val Thr Asn Val Val Arg Arg Asp Val Glu Lys
1940 1945 1950
Trp Gly Pro Phe Asp Leu Val Tyr Gly Ser Thr Gln Pro Leu Gly
1955 1960 1965
Ser Ser Cys Asp Arg Cys Pro Gly Trp Tyr Met Phe Gln Phe His
1970 1975 1980
Arg Ile Leu Gln Tyr Ala Leu Pro Arg Gln Glu Ser Gln Arg Pro
1985 1990 1995
Phe Phe Trp Ile Phe Met Asp Asn Leu Leu Leu Thr Glu Asp Asp
2000 2005 2010
Gln Glu Thr Thr Thr Arg Phe Leu Gln Thr Glu Ala Val Thr Leu
2015 2020 2025
Gln Asp Val Arg Gly Arg Asp Tyr Gln Asn Ala Met Arg Val Trp
2030 2035 2040
Ser Asn Ile Pro Gly Leu Lys Ser Lys His Ala Pro Leu Thr Pro
2045 2050 2055
Lys Glu Glu Glu Tyr Leu Gln Ala Gln Val Arg Ser Arg Ser Lys
2060 2065 2070
Leu Asp Ala Pro Lys Val Asp Leu Leu Val Lys Asn Cys Leu Leu
2075 2080 2085
Pro Leu Arg Glu Tyr Phe Lys Tyr Phe Ser Gln Asn Ser Leu Pro
2090 2095 2100
Leu Ser Arg Ala Asp Pro Lys Lys Lys Arg Lys Val Gly Ser Gly
2105 2110 2115
Ala Thr Asn Phe Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu
2120 2125 2130
Asn Pro Gly Pro Ser Glu Leu Ile Lys Glu Asn Met His Met Lys
2135 2140 2145
Leu Tyr Met Glu Gly Thr Val Asp Asn His His Phe Lys Cys Thr
2150 2155 2160
Ser Glu Gly Glu Gly Lys Pro Tyr Glu Gly Thr Gln Thr Met Arg
2165 2170 2175
Ile Lys Val Val Glu Gly Gly Pro Leu Pro Phe Ala Phe Asp Ile
2180 2185 2190
Leu Ala Thr Ser Phe Leu Tyr Gly Ser Lys Thr Phe Ile Asn His
2195 2200 2205
Thr Gln Gly Ile Pro Asp Phe Phe Lys Gln Ser Phe Pro Glu Gly
2210 2215 2220
Phe Thr Trp Glu Arg Val Thr Thr Tyr Glu Asp Gly Gly Val Leu
2225 2230 2235
Thr Ala Thr Gln Asp Thr Ser Leu Gln Asp Gly Cys Leu Ile Tyr
2240 2245 2250
Asn Val Lys Ile Arg Gly Val Asn Phe Thr Ser Asn Gly Pro Val
2255 2260 2265
Met Gln Lys Lys Thr Leu Gly Trp Glu Ala Phe Thr Glu Thr Leu
2270 2275 2280
Tyr Pro Ala Asp Gly Gly Leu Glu Gly Arg Asn Asp Met Ala Leu
2285 2290 2295
Lys Leu Val Gly Gly Ser His Leu Ile Ala Asn Ile Lys Thr Thr
2300 2305 2310
Tyr Arg Ser Lys Lys Pro Ala Lys Asn Leu Lys Met Pro Gly Val
2315 2320 2325
Tyr Tyr Val Asp Tyr Arg Leu Glu Arg Ile Lys Glu Ala Asn Asn
2330 2335 2340
Glu Thr Tyr Val Glu Gln His Glu Val Ala Val Ala Arg Tyr Cys
2345 2350 2355
Asp Leu Pro Ser Lys Leu Gly His Lys Leu Asn
2360 2365
<210> 14
<211> 2391
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетический полипептид
<400> 14
Asp Ala Lys Ser Leu Thr Ala Trp Ser Arg Thr Leu Val Thr Phe Lys
1. 5 10 15
Asp Val Phe Val Asp Phe Thr Arg Glu Glu Trp Lys Leu Leu Asp Thr
20 25 30
Ala Gln Gln Ile Val Tyr Arg Asn Val Met Leu Glu Asn Tyr Lys Asn
35 40 45
Leu Val Ser Leu Gly Tyr Gln Leu Thr Lys Pro Asp Val Ile Leu Arg
50 55 60
Leu Glu Lys Gly Glu Glu Pro Gly Gly Pro Ser Ser Gly Ala Pro Pro
65 70 75 80
Pro Ser Gly Gly Ser Pro Ala Gly Ser Pro Thr Ser Thr Glu Glu Gly
85 90 95
Thr Ser Glu Ser Ala Thr Pro Glu Ser Gly Pro Gly Thr Ser Thr Glu
100 105 110
Pro Ser Glu Gly Ser Ala Pro Gly Ser Pro Ala Gly Ser Pro Thr Ser
115 120 125
Thr Glu Glu Gly Thr Ser Thr Glu Pro Ser Glu Gly Ser Ala Pro Gly
130 135 140
Thr Ser Thr Glu Pro Ser Glu Met Asp Lys Lys Tyr Ser Ile Gly Leu
145 150 155 160
Ala Ile Gly Thr Asn Ser Val Gly Trp Ala Val Ile Thr Asp Glu Tyr
165 170 175
Lys Val Pro Ser Lys Lys Phe Lys Val Leu Gly Asn Thr Asp Arg His
180 185 190
Ser Ile Lys Lys Asn Leu Ile Gly Ala Leu Leu Phe Asp Ser Gly Glu
195 200 205
Thr Ala Glu Ala Thr Arg Leu Lys Arg Thr Ala Arg Arg Arg Tyr Thr
210 215 220
Arg Arg Lys Asn Arg Ile Cys Tyr Leu Gln Glu Ile Phe Ser Asn Glu
225 230 235 240
Met Ala Lys Val Asp Asp Ser Phe Phe His Arg Leu Glu Glu Ser Phe
245 250 255
Leu Val Glu Glu Asp Lys Lys His Glu Arg His Pro Ile Phe Gly Asn
260 265 270
Ile Val Asp Glu Val Ala Tyr His Glu Lys Tyr Pro Thr Ile Tyr His
275 280 285
Leu Arg Lys Lys Leu Val Asp Ser Thr Asp Lys Ala Asp Leu Arg Leu
290 295 300
Ile Tyr Leu Ala Leu Ala His Met Ile Lys Phe Arg Gly His Phe Leu
305 310 315 320
Ile Glu Gly Asp Leu Asn Pro Asp Asn Ser Asp Val Asp Lys Leu Phe
325 330 335
Ile Gln Leu Val Gln Thr Tyr Asn Gln Leu Phe Glu Glu Asn Pro Ile
340 345 350
Asn Ala Ser Gly Val Asp Ala Lys Ala Ile Leu Ser Ala Arg Leu Ser
355 360 365
Lys Ser Arg Arg Leu Glu Asn Leu Ile Ala Gln Leu Pro Gly Glu Lys
370 375 380
Lys Asn Gly Leu Phe Gly Asn Leu Ile Ala Leu Ser Leu Gly Leu Thr
385 390 395 400
Pro Asn Phe Lys Ser Asn Phe Asp Leu Ala Glu Asp Ala Lys Leu Gln
405 410 415
Leu Ser Lys Asp Thr Tyr Asp Asp Asp Leu Asp Asn Leu Leu Ala Gln
420 425 430
Ile Gly Asp Gln Tyr Ala Asp Leu Phe Leu Ala Ala Lys Asn Leu Ser
435 440 445
Asp Ala Ile Leu Leu Ser Asp Ile Leu Arg Val Asn Thr Glu Ile Thr
450 455 460
Lys Ala Pro Leu Ser Ala Ser Met Ile Lys Arg Tyr Asp Glu His His
465 470 475 480
Gln Asp Leu Thr Leu Leu Lys Ala Leu Val Arg Gln Gln Leu Pro Glu
485 490 495
Lys Tyr Lys Glu Ile Phe Phe Asp Gln Ser Lys Asn Gly Tyr Ala Gly
500 505 510
Tyr Ile Asp Gly Gly Ala Ser Gln Glu Glu Phe Tyr Lys Phe Ile Lys
515 520 525
Pro Ile Leu Glu Lys Met Asp Gly Thr Glu Glu Leu Leu Val Lys Leu
530 535 540
Asn Arg Glu Asp Leu Leu Arg Lys Gln Arg Thr Phe Asp Asn Gly Ser
545 550 555 560
Ile Pro His Gln Ile His Leu Gly Glu Leu His Ala Ile Leu Arg Arg
565 570 575
Gln Glu Asp Phe Tyr Pro Phe Leu Lys Asp Asn Arg Glu Lys Ile Glu
580 585 590
Lys Ile Leu Thr Phe Arg Ile Pro Tyr Tyr Val Gly Pro Leu Ala Arg
595 600 605
Gly Asn Ser Arg Phe Ala Trp Met Thr Arg Lys Ser Glu Glu Thr Ile
610 615 620
Thr Pro Trp Asn Phe Glu Glu Val Val Asp Lys Gly Ala Ser Ala Gln
625 630 635 640
Ser Phe Ile Glu Arg Met Thr Asn Phe Asp Lys Asn Leu Pro Asn Glu
645 650 655
Lys Val Leu Pro Lys His Ser Leu Leu Tyr Glu Tyr Phe Thr Val Tyr
660 665 670
Asn Glu Leu Thr Lys Val Lys Tyr Val Thr Glu Gly Met Arg Lys Pro
675 680 685
Ala Phe Leu Ser Gly Glu Gln Lys Lys Ala Ile Val Asp Leu Leu Phe
690 695 700
Lys Thr Asn Arg Lys Val Thr Val Lys Gln Leu Lys Glu Asp Tyr Phe
705 710 715 720
Lys Lys Ile Glu Cys Phe Asp Ser Val Glu Ile Ser Gly Val Glu Asp
725 730 735
Arg Phe Asn Ala Ser Leu Gly Thr Tyr His Asp Leu Leu Lys Ile Ile
740 745 750
Lys Asp Lys Asp Phe Leu Asp Asn Glu Glu Asn Glu Asp Ile Leu Glu
755 760 765
Asp Ile Val Leu Thr Leu Thr Leu Phe Glu Asp Arg Glu Met Ile Glu
770 775 780
Glu Arg Leu Lys Thr Tyr Ala His Leu Phe Asp Asp Lys Val Met Lys
785 790 795 800
Gln Leu Lys Arg Arg Arg Tyr Thr Gly Trp Gly Arg Leu Ser Arg Lys
805 810 815
Leu Ile Asn Gly Ile Arg Asp Lys Gln Ser Gly Lys Thr Ile Leu Asp
820 825 830
Phe Leu Lys Ser Asp Gly Phe Ala Asn Arg Asn Phe Met Gln Leu Ile
835 840 845
His Asp Asp Ser Leu Thr Phe Lys Glu Asp Ile Gln Lys Ala Gln Val
850 855 860
Ser Gly Gln Gly Asp Ser Leu His Glu His Ile Ala Asn Leu Ala Gly
865 870 875 880
Ser Pro Ala Ile Lys Lys Gly Ile Leu Gln Thr Val Lys Val Val Asp
885 890 895
Glu Leu Val Lys Val Met Gly Arg His Lys Pro Glu Asn Ile Val Ile
900 905 910
Glu Met Ala Arg Glu Asn Gln Thr Thr Gln Lys Gly Gln Lys Asn Ser
915 920 925
Arg Glu Arg Met Lys Arg Ile Glu Glu Gly Ile Lys Glu Leu Gly Ser
930 935 940
Gln Ile Leu Lys Glu His Pro Val Glu Asn Thr Gln Leu Gln Asn Glu
945 950 955 960
Lys Leu Tyr Leu Tyr Tyr Leu Gln Asn Gly Arg Asp Met Tyr Val Asp
965 970 975
Gln Glu Leu Asp Ile Asn Arg Leu Ser Asp Tyr Asp Val Asp Ala Ile
980 985 990
Val Pro Gln Ser Phe Leu Lys Asp Asp Ser Ile Asp Asn Lys Val Leu
995 1000 1005
Thr Arg Ser Asp Lys Asn Arg Gly Lys Ser Asp Asn Val Pro Ser
1010 1015 1020
Glu Glu Val Val Lys Lys Met Lys Asn Tyr Trp Arg Gln Leu Leu
1025 1030 1035
Asn Ala Lys Leu Ile Thr Gln Arg Lys Phe Asp Asn Leu Thr Lys
1040 1045 1050
Ala Glu Arg Gly Gly Leu Ser Glu Leu Asp Lys Ala Gly Phe Ile
1055 1060 1065
Lys Arg Gln Leu Val Glu Thr Arg Gln Ile Thr Lys His Val Ala
1070 1075 1080
Gln Ile Leu Asp Ser Arg Met Asn Thr Lys Tyr Asp Glu Asn Asp
1085 1090 1095
Lys Leu Ile Arg Glu Val Lys Val Ile Thr Leu Lys Ser Lys Leu
1100 1105 1110
Val Ser Asp Phe Arg Lys Asp Phe Gln Phe Tyr Lys Val Arg Glu
1115 1120 1125
Ile Asn Asn Tyr His His Ala His Asp Ala Tyr Leu Asn Ala Val
1130 1135 1140
Val Gly Thr Ala Leu Ile Lys Lys Tyr Pro Lys Leu Glu Ser Glu
1145 1150 1155
Phe Val Tyr Gly Asp Tyr Lys Val Tyr Asp Val Arg Lys Met Ile
1160 1165 1170
Ala Lys Ser Glu Gln Glu Ile Gly Lys Ala Thr Ala Lys Tyr Phe
1175 1180 1185
Phe Tyr Ser Asn Ile Met Asn Phe Phe Lys Thr Glu Ile Thr Leu
1190 1195 1200
Ala Asn Gly Glu Ile Arg Lys Arg Pro Leu Ile Glu Thr Asn Gly
1205 1210 1215
Glu Thr Gly Glu Ile Val Trp Asp Lys Gly Arg Asp Phe Ala Thr
1220 1225 1230
Val Arg Lys Val Leu Ser Met Pro Gln Val Asn Ile Val Lys Lys
1235 1240 1245
Thr Glu Val Gln Thr Gly Gly Phe Ser Lys Glu Ser Ile Leu Pro
1250 1255 1260
Lys Arg Asn Ser Asp Lys Leu Ile Ala Arg Lys Lys Asp Trp Asp
1265 1270 1275
Pro Lys Lys Tyr Gly Gly Phe Asp Ser Pro Thr Val Ala Tyr Ser
1280 1285 1290
Val Leu Val Val Ala Lys Val Glu Lys Gly Lys Ser Lys Lys Leu
1295 1300 1305
Lys Ser Val Lys Glu Leu Leu Gly Ile Thr Ile Met Glu Arg Ser
1310 1315 1320
Ser Phe Glu Lys Asn Pro Ile Asp Phe Leu Glu Ala Lys Gly Tyr
1325 1330 1335
Lys Glu Val Lys Lys Asp Leu Ile Ile Lys Leu Pro Lys Tyr Ser
1340 1345 1350
Leu Phe Glu Leu Glu Asn Gly Arg Lys Arg Met Leu Ala Ser Ala
1355 1360 1365
Gly Glu Leu Gln Lys Gly Asn Glu Leu Ala Leu Pro Ser Lys Tyr
1370 1375 1380
Val Asn Phe Leu Tyr Leu Ala Ser His Tyr Glu Lys Leu Lys Gly
1385 1390 1395
Ser Pro Glu Asp Asn Glu Gln Lys Gln Leu Phe Val Glu Gln His
1400 1405 1410
Lys His Tyr Leu Asp Glu Ile Ile Glu Gln Ile Ser Glu Phe Ser
1415 1420 1425
Lys Arg Val Ile Leu Ala Asp Ala Asn Leu Asp Lys Val Leu Ser
1430 1435 1440
Ala Tyr Asn Lys His Arg Asp Lys Pro Ile Arg Glu Gln Ala Glu
1445 1450 1455
Asn Ile Ile His Leu Phe Thr Leu Thr Asn Leu Gly Ala Pro Ala
1460 1465 1470
Ala Phe Lys Tyr Phe Asp Thr Thr Ile Asp Arg Lys Arg Tyr Thr
1475 1480 1485
Ser Thr Lys Glu Val Leu Asp Ala Thr Leu Ile His Gln Ser Ile
1490 1495 1500
Thr Gly Leu Tyr Glu Thr Arg Ile Asp Leu Ser Gln Leu Gly Gly
1505 1510 1515
Asp Ser Arg Ala Asp Tyr Pro Tyr Asp Val Pro Asp Tyr Ala Ser
1520 1525 1530
Gly Ser Pro Lys Lys Lys Arg Lys Val Ser Pro Gly Ser Gly Ser
1535 1540 1545
Glu Thr Pro Gly Thr Ser Glu Ser Ala Thr Pro Glu Ser Asn His
1550 1555 1560
Asp Gln Glu Phe Asp Pro Pro Lys Val Tyr Pro Pro Val Pro Ala
1565 1570 1575
Glu Lys Arg Lys Pro Ile Arg Val Leu Ser Leu Phe Asp Gly Ile
1580 1585 1590
Ala Thr Gly Leu Leu Val Leu Lys Asp Leu Gly Ile Gln Val Asp
1595 1600 1605
Arg Tyr Ile Ala Ser Glu Val Cys Glu Asp Ser Ile Thr Val Gly
1610 1615 1620
Met Val Arg His Gln Gly Lys Ile Met Tyr Val Gly Asp Val Arg
1625 1630 1635
Ser Val Thr Gln Lys His Ile Gln Glu Trp Gly Pro Phe Asp Leu
1640 1645 1650
Val Ile Gly Gly Ser Pro Cys Asn Asp Leu Ser Ile Val Asn Pro
1655 1660 1665
Ala Arg Lys Gly Leu Tyr Glu Gly Thr Gly Arg Leu Phe Phe Glu
1670 1675 1680
Phe Tyr Arg Leu Leu His Asp Ala Arg Pro Lys Glu Gly Asp Asp
1685 1690 1695
Arg Pro Phe Phe Trp Leu Phe Glu Asn Val Val Ala Met Gly Val
1700 1705 1710
Ser Asp Lys Arg Asp Ile Ser Arg Phe Leu Glu Ser Asn Pro Val
1715 1720 1725
Met Ile Asp Ala Lys Glu Val Ser Ala Ala His Arg Ala Arg Tyr
1730 1735 1740
Phe Trp Gly Asn Leu Pro Gly Met Asn Arg Pro Leu Ala Ser Thr
1745 1750 1755
Val Asn Asp Lys Leu Glu Leu Gln Glu Cys Leu Glu His Gly Arg
1760 1765 1770
Ile Ala Lys Phe Ser Lys Val Arg Thr Ile Thr Thr Arg Ser Asn
1775 1780 1785
Ser Ile Lys Gln Gly Lys Asp Gln His Phe Pro Val Phe Met Asn
1790 1795 1800
Glu Lys Glu Asp Ile Leu Trp Cys Thr Glu Met Glu Arg Val Phe
1805 1810 1815
Gly Phe Pro Val His Tyr Thr Asp Val Ser Asn Met Ser Arg Leu
1820 1825 1830
Ala Arg Gln Arg Leu Leu Gly Arg Ser Trp Ser Val Pro Val Ile
1835 1840 1845
Arg His Leu Phe Ala Pro Leu Lys Glu Tyr Phe Ala Cys Val Ser
1850 1855 1860
Ser Gly Asn Ser Asn Ala Asn Ser Arg Gly Pro Ser Phe Ser Ser
1865 1870 1875
Gly Leu Val Pro Leu Ser Leu Arg Gly Ser His Met Gly Pro Met
1880 1885 1890
Glu Ile Tyr Lys Thr Val Ser Ala Trp Lys Arg Gln Pro Val Arg
1895 1900 1905
Val Leu Ser Leu Phe Arg Asn Ile Asp Lys Val Leu Lys Ser Leu
1910 1915 1920
Gly Phe Leu Glu Ser Gly Ser Gly Ser Gly Gly Gly Thr Leu Lys
1925 1930 1935
Tyr Val Glu Asp Val Thr Asn Val Val Arg Arg Asp Val Glu Lys
1940 1945 1950
Trp Gly Pro Phe Asp Leu Val Tyr Gly Ser Thr Gln Pro Leu Gly
1955 1960 1965
Ser Ser Cys Asp Arg Cys Pro Gly Trp Tyr Met Phe Gln Phe His
1970 1975 1980
Arg Ile Leu Gln Tyr Ala Leu Pro Arg Gln Glu Ser Gln Arg Pro
1985 1990 1995
Phe Phe Trp Ile Phe Met Asp Asn Leu Leu Leu Thr Glu Asp Asp
2000 2005 2010
Gln Glu Thr Thr Thr Arg Phe Leu Gln Thr Glu Ala Val Thr Leu
2015 2020 2025
Gln Asp Val Arg Gly Arg Asp Tyr Gln Asn Ala Met Arg Val Trp
2030 2035 2040
Ser Asn Ile Pro Gly Leu Lys Ser Lys His Ala Pro Leu Thr Pro
2045 2050 2055
Lys Glu Glu Glu Tyr Leu Gln Ala Gln Val Arg Ser Arg Ser Lys
2060 2065 2070
Leu Asp Ala Pro Lys Val Asp Leu Leu Val Lys Asn Cys Leu Leu
2075 2080 2085
Pro Leu Arg Glu Tyr Phe Lys Tyr Phe Ser Gln Asn Ser Leu Pro
2090 2095 2100
Leu Ser Arg Ala Asp Pro Lys Lys Lys Arg Lys Val Gly Ser Gly
2105 2110 2115
Ala Thr Asn Phe Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu
2120 2125 2130
Asn Pro Gly Pro Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu Lys
2135 2140 2145
Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Ser Glu Leu Ile
2150 2155 2160
Lys Glu Asn Met His Met Lys Leu Tyr Met Glu Gly Thr Val Asp
2165 2170 2175
Asn His His Phe Lys Cys Thr Ser Glu Gly Glu Gly Lys Pro Tyr
2180 2185 2190
Glu Gly Thr Gln Thr Met Arg Ile Lys Val Val Glu Gly Gly Pro
2195 2200 2205
Leu Pro Phe Ala Phe Asp Ile Leu Ala Thr Ser Phe Leu Tyr Gly
2210 2215 2220
Ser Lys Thr Phe Ile Asn His Thr Gln Gly Ile Pro Asp Phe Phe
2225 2230 2235
Lys Gln Ser Phe Pro Glu Gly Phe Thr Trp Glu Arg Val Thr Thr
2240 2245 2250
Tyr Glu Asp Gly Gly Val Leu Thr Ala Thr Gln Asp Thr Ser Leu
2255 2260 2265
Gln Asp Gly Cys Leu Ile Tyr Asn Val Lys Ile Arg Gly Val Asn
2270 2275 2280
Phe Thr Ser Asn Gly Pro Val Met Gln Lys Lys Thr Leu Gly Trp
2285 2290 2295
Glu Ala Phe Thr Glu Thr Leu Tyr Pro Ala Asp Gly Gly Leu Glu
2300 2305 2310
Gly Arg Asn Asp Met Ala Leu Lys Leu Val Gly Gly Ser His Leu
2315 2320 2325
Ile Ala Asn Ile Lys Thr Thr Tyr Arg Ser Lys Lys Pro Ala Lys
2330 2335 2340
Asn Leu Lys Met Pro Gly Val Tyr Tyr Val Asp Tyr Arg Leu Glu
2345 2350 2355
Arg Ile Lys Glu Ala Asn Asn Glu Thr Tyr Val Glu Gln His Glu
2360 2365 2370
Val Ala Val Ala Arg Tyr Cys Asp Leu Pro Ser Lys Leu Gly His
2375 2380 2385
Lys Leu Asn
2390
<210> 15
<211> 2345
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетический полипептид
<400> 15
Asn His Asp Gln Glu Phe Asp Pro Pro Lys Val Tyr Pro Pro Val Pro
1. 5 10 15
Ala Glu Lys Arg Lys Pro Ile Arg Val Leu Ser Leu Phe Asp Gly Ile
20 25 30
Ala Thr Gly Leu Leu Val Leu Lys Asp Leu Gly Ile Gln Val Asp Arg
35 40 45
Tyr Ile Ala Ser Glu Val Cys Glu Asp Ser Ile Thr Val Gly Met Val
50 55 60
Arg His Gln Gly Lys Ile Met Tyr Val Gly Asp Val Arg Ser Val Thr
65 70 75 80
Gln Lys His Ile Gln Glu Trp Gly Pro Phe Asp Leu Val Ile Gly Gly
85 90 95
Ser Pro Cys Asn Asp Leu Ser Ile Val Asn Pro Ala Arg Lys Gly Leu
100 105 110
Tyr Glu Gly Thr Gly Arg Leu Phe Phe Glu Phe Tyr Arg Leu Leu His
115 120 125
Asp Ala Arg Pro Lys Glu Gly Asp Asp Arg Pro Phe Phe Trp Leu Phe
130 135 140
Glu Asn Val Val Ala Met Gly Val Ser Asp Lys Arg Asp Ile Ser Arg
145 150 155 160
Phe Leu Glu Ser Asn Pro Val Met Ile Asp Ala Lys Glu Val Ser Ala
165 170 175
Ala His Arg Ala Arg Tyr Phe Trp Gly Asn Leu Pro Gly Met Asn Arg
180 185 190
Pro Leu Ala Ser Thr Val Asn Asp Lys Leu Glu Leu Gln Glu Cys Leu
195 200 205
Glu His Gly Arg Ile Ala Lys Phe Ser Lys Val Arg Thr Ile Thr Thr
210 215 220
Arg Ser Asn Ser Ile Lys Gln Gly Lys Asp Gln His Phe Pro Val Phe
225 230 235 240
Met Asn Glu Lys Glu Asp Ile Leu Trp Cys Thr Glu Met Glu Arg Val
245 250 255
Phe Gly Phe Pro Val His Tyr Thr Asp Val Ser Asn Met Ser Arg Leu
260 265 270
Ala Arg Gln Arg Leu Leu Gly Arg Ser Trp Ser Val Pro Val Ile Arg
275 280 285
His Leu Phe Ala Pro Leu Lys Glu Tyr Phe Ala Cys Val Ser Ser Gly
290 295 300
Asn Ser Asn Ala Asn Ser Arg Gly Pro Ser Phe Ser Ser Gly Leu Val
305 310 315 320
Pro Leu Ser Leu Arg Gly Ser His Met Gly Pro Met Glu Ile Tyr Lys
325 330 335
Thr Val Ser Ala Trp Lys Arg Gln Pro Val Arg Val Leu Ser Leu Phe
340 345 350
Arg Asn Ile Asp Lys Val Leu Lys Ser Leu Gly Phe Leu Glu Ser Gly
355 360 365
Ser Gly Ser Gly Gly Gly Thr Leu Lys Tyr Val Glu Asp Val Thr Asn
370 375 380
Val Val Arg Arg Asp Val Glu Lys Trp Gly Pro Phe Asp Leu Val Tyr
385 390 395 400
Gly Ser Thr Gln Pro Leu Gly Ser Ser Cys Asp Arg Cys Pro Gly Trp
405 410 415
Tyr Met Phe Gln Phe His Arg Ile Leu Gln Tyr Ala Leu Pro Arg Gln
420 425 430
Glu Ser Gln Arg Pro Phe Phe Trp Ile Phe Met Asp Asn Leu Leu Leu
435 440 445
Thr Glu Asp Asp Gln Glu Thr Thr Thr Arg Phe Leu Gln Thr Glu Ala
450 455 460
Val Thr Leu Gln Asp Val Arg Gly Arg Asp Tyr Gln Asn Ala Met Arg
465 470 475 480
Val Trp Ser Asn Ile Pro Gly Leu Lys Ser Lys His Ala Pro Leu Thr
485 490 495
Pro Lys Glu Glu Glu Tyr Leu Gln Ala Gln Val Arg Ser Arg Ser Lys
500 505 510
Leu Asp Ala Pro Lys Val Asp Leu Leu Val Lys Asn Cys Leu Leu Pro
515 520 525
Leu Arg Glu Tyr Phe Lys Tyr Phe Ser Gln Asn Ser Leu Pro Leu Gly
530 535 540
Gly Pro Ser Ser Gly Ala Pro Pro Pro Ser Gly Gly Ser Pro Ala Gly
545 550 555 560
Ser Pro Thr Ser Thr Glu Glu Gly Thr Ser Glu Ser Ala Thr Pro Glu
565 570 575
Ser Gly Pro Gly Thr Ser Thr Glu Pro Ser Glu Gly Ser Ala Pro Gly
580 585 590
Ser Pro Ala Gly Ser Pro Thr Ser Thr Glu Glu Gly Thr Ser Thr Glu
595 600 605
Pro Ser Glu Gly Ser Ala Pro Gly Thr Ser Thr Glu Pro Ser Glu Met
610 615 620
Asp Lys Lys Tyr Ser Ile Gly Leu Ala Ile Gly Thr Asn Ser Val Gly
625 630 635 640
Trp Ala Val Ile Thr Asp Glu Tyr Lys Val Pro Ser Lys Lys Phe Lys
645 650 655
Val Leu Gly Asn Thr Asp Arg His Ser Ile Lys Lys Asn Leu Ile Gly
660 665 670
Ala Leu Leu Phe Asp Ser Gly Glu Thr Ala Glu Ala Thr Arg Leu Lys
675 680 685
Arg Thr Ala Arg Arg Arg Tyr Thr Arg Arg Lys Asn Arg Ile Cys Tyr
690 695 700
Leu Gln Glu Ile Phe Ser Asn Glu Met Ala Lys Val Asp Asp Ser Phe
705 710 715 720
Phe His Arg Leu Glu Glu Ser Phe Leu Val Glu Glu Asp Lys Lys His
725 730 735
Glu Arg His Pro Ile Phe Gly Asn Ile Val Asp Glu Val Ala Tyr His
740 745 750
Glu Lys Tyr Pro Thr Ile Tyr His Leu Arg Lys Lys Leu Val Asp Ser
755 760 765
Thr Asp Lys Ala Asp Leu Arg Leu Ile Tyr Leu Ala Leu Ala His Met
770 775 780
Ile Lys Phe Arg Gly His Phe Leu Ile Glu Gly Asp Leu Asn Pro Asp
785 790 795 800
Asn Ser Asp Val Asp Lys Leu Phe Ile Gln Leu Val Gln Thr Tyr Asn
805 810 815
Gln Leu Phe Glu Glu Asn Pro Ile Asn Ala Ser Gly Val Asp Ala Lys
820 825 830
Ala Ile Leu Ser Ala Arg Leu Ser Lys Ser Arg Arg Leu Glu Asn Leu
835 840 845
Ile Ala Gln Leu Pro Gly Glu Lys Lys Asn Gly Leu Phe Gly Asn Leu
850 855 860
Ile Ala Leu Ser Leu Gly Leu Thr Pro Asn Phe Lys Ser Asn Phe Asp
865 870 875 880
Leu Ala Glu Asp Ala Lys Leu Gln Leu Ser Lys Asp Thr Tyr Asp Asp
885 890 895
Asp Leu Asp Asn Leu Leu Ala Gln Ile Gly Asp Gln Tyr Ala Asp Leu
900 905 910
Phe Leu Ala Ala Lys Asn Leu Ser Asp Ala Ile Leu Leu Ser Asp Ile
915 920 925
Leu Arg Val Asn Thr Glu Ile Thr Lys Ala Pro Leu Ser Ala Ser Met
930 935 940
Ile Lys Arg Tyr Asp Glu His His Gln Asp Leu Thr Leu Leu Lys Ala
945 950 955 960
Leu Val Arg Gln Gln Leu Pro Glu Lys Tyr Lys Glu Ile Phe Phe Asp
965 970 975
Gln Ser Lys Asn Gly Tyr Ala Gly Tyr Ile Asp Gly Gly Ala Ser Gln
980 985 990
Glu Glu Phe Tyr Lys Phe Ile Lys Pro Ile Leu Glu Lys Met Asp Gly
995 1000 1005
Thr Glu Glu Leu Leu Val Lys Leu Asn Arg Glu Asp Leu Leu Arg
1010 1015 1020
Lys Gln Arg Thr Phe Asp Asn Gly Ser Ile Pro His Gln Ile His
1025 1030 1035
Leu Gly Glu Leu His Ala Ile Leu Arg Arg Gln Glu Asp Phe Tyr
1040 1045 1050
Pro Phe Leu Lys Asp Asn Arg Glu Lys Ile Glu Lys Ile Leu Thr
1055 1060 1065
Phe Arg Ile Pro Tyr Tyr Val Gly Pro Leu Ala Arg Gly Asn Ser
1070 1075 1080
Arg Phe Ala Trp Met Thr Arg Lys Ser Glu Glu Thr Ile Thr Pro
1085 1090 1095
Trp Asn Phe Glu Glu Val Val Asp Lys Gly Ala Ser Ala Gln Ser
1100 1105 1110
Phe Ile Glu Arg Met Thr Asn Phe Asp Lys Asn Leu Pro Asn Glu
1115 1120 1125
Lys Val Leu Pro Lys His Ser Leu Leu Tyr Glu Tyr Phe Thr Val
1130 1135 1140
Tyr Asn Glu Leu Thr Lys Val Lys Tyr Val Thr Glu Gly Met Arg
1145 1150 1155
Lys Pro Ala Phe Leu Ser Gly Glu Gln Lys Lys Ala Ile Val Asp
1160 1165 1170
Leu Leu Phe Lys Thr Asn Arg Lys Val Thr Val Lys Gln Leu Lys
1175 1180 1185
Glu Asp Tyr Phe Lys Lys Ile Glu Cys Phe Asp Ser Val Glu Ile
1190 1195 1200
Ser Gly Val Glu Asp Arg Phe Asn Ala Ser Leu Gly Thr Tyr His
1205 1210 1215
Asp Leu Leu Lys Ile Ile Lys Asp Lys Asp Phe Leu Asp Asn Glu
1220 1225 1230
Glu Asn Glu Asp Ile Leu Glu Asp Ile Val Leu Thr Leu Thr Leu
1235 1240 1245
Phe Glu Asp Arg Glu Met Ile Glu Glu Arg Leu Lys Thr Tyr Ala
1250 1255 1260
His Leu Phe Asp Asp Lys Val Met Lys Gln Leu Lys Arg Arg Arg
1265 1270 1275
Tyr Thr Gly Trp Gly Arg Leu Ser Arg Lys Leu Ile Asn Gly Ile
1280 1285 1290
Arg Asp Lys Gln Ser Gly Lys Thr Ile Leu Asp Phe Leu Lys Ser
1295 1300 1305
Asp Gly Phe Ala Asn Arg Asn Phe Met Gln Leu Ile His Asp Asp
1310 1315 1320
Ser Leu Thr Phe Lys Glu Asp Ile Gln Lys Ala Gln Val Ser Gly
1325 1330 1335
Gln Gly Asp Ser Leu His Glu His Ile Ala Asn Leu Ala Gly Ser
1340 1345 1350
Pro Ala Ile Lys Lys Gly Ile Leu Gln Thr Val Lys Val Val Asp
1355 1360 1365
Glu Leu Val Lys Val Met Gly Arg His Lys Pro Glu Asn Ile Val
1370 1375 1380
Ile Glu Met Ala Arg Glu Asn Gln Thr Thr Gln Lys Gly Gln Lys
1385 1390 1395
Asn Ser Arg Glu Arg Met Lys Arg Ile Glu Glu Gly Ile Lys Glu
1400 1405 1410
Leu Gly Ser Gln Ile Leu Lys Glu His Pro Val Glu Asn Thr Gln
1415 1420 1425
Leu Gln Asn Glu Lys Leu Tyr Leu Tyr Tyr Leu Gln Asn Gly Arg
1430 1435 1440
Asp Met Tyr Val Asp Gln Glu Leu Asp Ile Asn Arg Leu Ser Asp
1445 1450 1455
Tyr Asp Val Asp Ala Ile Val Pro Gln Ser Phe Leu Lys Asp Asp
1460 1465 1470
Ser Ile Asp Asn Lys Val Leu Thr Arg Ser Asp Lys Asn Arg Gly
1475 1480 1485
Lys Ser Asp Asn Val Pro Ser Glu Glu Val Val Lys Lys Met Lys
1490 1495 1500
Asn Tyr Trp Arg Gln Leu Leu Asn Ala Lys Leu Ile Thr Gln Arg
1505 1510 1515
Lys Phe Asp Asn Leu Thr Lys Ala Glu Arg Gly Gly Leu Ser Glu
1520 1525 1530
Leu Asp Lys Ala Gly Phe Ile Lys Arg Gln Leu Val Glu Thr Arg
1535 1540 1545
Gln Ile Thr Lys His Val Ala Gln Ile Leu Asp Ser Arg Met Asn
1550 1555 1560
Thr Lys Tyr Asp Glu Asn Asp Lys Leu Ile Arg Glu Val Lys Val
1565 1570 1575
Ile Thr Leu Lys Ser Lys Leu Val Ser Asp Phe Arg Lys Asp Phe
1580 1585 1590
Gln Phe Tyr Lys Val Arg Glu Ile Asn Asn Tyr His His Ala His
1595 1600 1605
Asp Ala Tyr Leu Asn Ala Val Val Gly Thr Ala Leu Ile Lys Lys
1610 1615 1620
Tyr Pro Lys Leu Glu Ser Glu Phe Val Tyr Gly Asp Tyr Lys Val
1625 1630 1635
Tyr Asp Val Arg Lys Met Ile Ala Lys Ser Glu Gln Glu Ile Gly
1640 1645 1650
Lys Ala Thr Ala Lys Tyr Phe Phe Tyr Ser Asn Ile Met Asn Phe
1655 1660 1665
Phe Lys Thr Glu Ile Thr Leu Ala Asn Gly Glu Ile Arg Lys Arg
1670 1675 1680
Pro Leu Ile Glu Thr Asn Gly Glu Thr Gly Glu Ile Val Trp Asp
1685 1690 1695
Lys Gly Arg Asp Phe Ala Thr Val Arg Lys Val Leu Ser Met Pro
1700 1705 1710
Gln Val Asn Ile Val Lys Lys Thr Glu Val Gln Thr Gly Gly Phe
1715 1720 1725
Ser Lys Glu Ser Ile Leu Pro Lys Arg Asn Ser Asp Lys Leu Ile
1730 1735 1740
Ala Arg Lys Lys Asp Trp Asp Pro Lys Lys Tyr Gly Gly Phe Asp
1745 1750 1755
Ser Pro Thr Val Ala Tyr Ser Val Leu Val Val Ala Lys Val Glu
1760 1765 1770
Lys Gly Lys Ser Lys Lys Leu Lys Ser Val Lys Glu Leu Leu Gly
1775 1780 1785
Ile Thr Ile Met Glu Arg Ser Ser Phe Glu Lys Asn Pro Ile Asp
1790 1795 1800
Phe Leu Glu Ala Lys Gly Tyr Lys Glu Val Lys Lys Asp Leu Ile
1805 1810 1815
Ile Lys Leu Pro Lys Tyr Ser Leu Phe Glu Leu Glu Asn Gly Arg
1820 1825 1830
Lys Arg Met Leu Ala Ser Ala Gly Glu Leu Gln Lys Gly Asn Glu
1835 1840 1845
Leu Ala Leu Pro Ser Lys Tyr Val Asn Phe Leu Tyr Leu Ala Ser
1850 1855 1860
His Tyr Glu Lys Leu Lys Gly Ser Pro Glu Asp Asn Glu Gln Lys
1865 1870 1875
Gln Leu Phe Val Glu Gln His Lys His Tyr Leu Asp Glu Ile Ile
1880 1885 1890
Glu Gln Ile Ser Glu Phe Ser Lys Arg Val Ile Leu Ala Asp Ala
1895 1900 1905
Asn Leu Asp Lys Val Leu Ser Ala Tyr Asn Lys His Arg Asp Lys
1910 1915 1920
Pro Ile Arg Glu Gln Ala Glu Asn Ile Ile His Leu Phe Thr Leu
1925 1930 1935
Thr Asn Leu Gly Ala Pro Ala Ala Phe Lys Tyr Phe Asp Thr Thr
1940 1945 1950
Ile Asp Arg Lys Arg Tyr Thr Ser Thr Lys Glu Val Leu Asp Ala
1955 1960 1965
Thr Leu Ile His Gln Ser Ile Thr Gly Leu Tyr Glu Thr Arg Ile
1970 1975 1980
Asp Leu Ser Gln Leu Gly Gly Asp Ala Tyr Pro Tyr Asp Val Pro
1985 1990 1995
Asp Tyr Ala Ser Leu Gly Ser Gly Ser Pro Lys Lys Lys Arg Lys
2000 2005 2010
Val Glu Asp Pro Lys Lys Lys Arg Lys Val Asp Gly Ile Gly Ser
2015 2020 2025
Gly Ser Asn Gly Ser Ser Gly Ser Ser Glu Leu Ile Lys Glu Asn
2030 2035 2040
Met His Met Lys Leu Tyr Met Glu Gly Thr Val Asp Asn His His
2045 2050 2055
Phe Lys Cys Thr Ser Glu Gly Glu Gly Lys Pro Tyr Glu Gly Thr
2060 2065 2070
Gln Thr Met Arg Ile Lys Val Val Glu Gly Gly Pro Leu Pro Phe
2075 2080 2085
Ala Phe Asp Ile Leu Ala Thr Ser Phe Leu Tyr Gly Ser Lys Thr
2090 2095 2100
Phe Ile Asn His Thr Gln Gly Ile Pro Asp Phe Phe Lys Gln Ser
2105 2110 2115
Phe Pro Glu Gly Phe Thr Trp Glu Arg Val Thr Thr Tyr Glu Asp
2120 2125 2130
Gly Gly Val Leu Thr Ala Thr Gln Asp Thr Ser Leu Gln Asp Gly
2135 2140 2145
Cys Leu Ile Tyr Asn Val Lys Ile Arg Gly Val Asn Phe Thr Ser
2150 2155 2160
Asn Gly Pro Val Met Gln Lys Lys Thr Leu Gly Trp Glu Ala Phe
2165 2170 2175
Thr Glu Thr Leu Tyr Pro Ala Asp Gly Gly Leu Glu Gly Arg Asn
2180 2185 2190
Asp Met Ala Leu Lys Leu Val Gly Gly Ser His Leu Ile Ala Asn
2195 2200 2205
Ile Lys Thr Thr Tyr Arg Ser Lys Lys Pro Ala Lys Asn Leu Lys
2210 2215 2220
Met Pro Gly Val Tyr Tyr Val Asp Tyr Arg Leu Glu Arg Ile Lys
2225 2230 2235
Glu Ala Asn Asn Glu Thr Tyr Val Glu Gln His Glu Val Ala Val
2240 2245 2250
Ala Arg Tyr Cys Asp Leu Pro Ser Lys Leu Gly His Lys Leu Asn
2255 2260 2265
Gly Gly Gly Gly Gly Met Asp Ala Lys Ser Leu Thr Ala Trp Ser
2270 2275 2280
Arg Thr Leu Val Thr Phe Lys Asp Val Phe Val Asp Phe Thr Arg
2285 2290 2295
Glu Glu Trp Lys Leu Leu Asp Thr Ala Gln Gln Ile Val Tyr Arg
2300 2305 2310
Asn Val Met Leu Glu Asn Tyr Lys Asn Leu Val Ser Leu Gly Tyr
2315 2320 2325
Gln Leu Thr Lys Pro Asp Val Ile Leu Arg Leu Glu Lys Gly Glu
2330 2335 2340
Glu Pro
2345
<210> 16
<211> 71
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетический полипептид
<400> 16
Asp Ala Lys Ser Leu Thr Ala Trp Ser Arg Thr Leu Val Thr Phe Lys
1. 5 10 15
Asp Val Phe Val Asp Phe Thr Arg Glu Glu Trp Lys Leu Leu Asp Thr
20 25 30
Ala Gln Gln Ile Val Tyr Arg Asn Val Met Leu Glu Asn Tyr Lys Asn
35 40 45
Leu Val Ser Leu Gly Tyr Gln Leu Thr Lys Pro Asp Val Ile Leu Arg
50 55 60
Leu Glu Lys Gly Glu Glu Pro
65 70
<210> 17
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетический полипептид
<400> 17
Gly Gly Ser Gly Gly Gly Ser
1. 5
<210> 18
<211> 3
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетический полипептид
<400> 18
Ser Gly Ser
1
<210> 19
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетический полипептид
<400> 19
Glu Ala Ser Gly Ser Gly Arg Ala Ser Pro Gly Ile Pro Gly Ser Thr
1. 5 10 15
Arg
<210> 20
<211> 4
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетический полипептид
<400> 20
Ser Arg Ala Asp
1
<210> 21
<211> 3
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетический полипептид
<400> 21
Gly Ser Gly
1
<210> 22
<211> 3
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетический полипептид
<400> 22
Ser Pro Gly
1
<210> 23
<211> 1368
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетический полипептид
<400> 23
Met Asp Lys Lys Tyr Ser Ile Gly Leu Ala Ile Gly Thr Asn Ser Val
1. 5 10 15
Gly Trp Ala Val Ile Thr Asp Glu Tyr Lys Val Pro Ser Lys Lys Phe
20 25 30
Lys Val Leu Gly Asn Thr Asp Arg His Ser Ile Lys Lys Asn Leu Ile
35 40 45
Gly Ala Leu Leu Phe Asp Ser Gly Glu Thr Ala Glu Ala Thr Arg Leu
50 55 60
Lys Arg Thr Ala Arg Arg Arg Tyr Thr Arg Arg Lys Asn Arg Ile Cys
65 70 75 80
Tyr Leu Gln Glu Ile Phe Ser Asn Glu Met Ala Lys Val Asp Asp Ser
85 90 95
Phe Phe His Arg Leu Glu Glu Ser Phe Leu Val Glu Glu Asp Lys Lys
100 105 110
His Glu Arg His Pro Ile Phe Gly Asn Ile Val Asp Glu Val Ala Tyr
115 120 125
His Glu Lys Tyr Pro Thr Ile Tyr His Leu Arg Lys Lys Leu Val Asp
130 135 140
Ser Thr Asp Lys Ala Asp Leu Arg Leu Ile Tyr Leu Ala Leu Ala His
145 150 155 160
Met Ile Lys Phe Arg Gly His Phe Leu Ile Glu Gly Asp Leu Asn Pro
165 170 175
Asp Asn Ser Asp Val Asp Lys Leu Phe Ile Gln Leu Val Gln Thr Tyr
180 185 190
Asn Gln Leu Phe Glu Glu Asn Pro Ile Asn Ala Ser Gly Val Asp Ala
195 200 205
Lys Ala Ile Leu Ser Ala Arg Leu Ser Lys Ser Arg Arg Leu Glu Asn
210 215 220
Leu Ile Ala Gln Leu Pro Gly Glu Lys Lys Asn Gly Leu Phe Gly Asn
225 230 235 240
Leu Ile Ala Leu Ser Leu Gly Leu Thr Pro Asn Phe Lys Ser Asn Phe
245 250 255
Asp Leu Ala Glu Asp Ala Lys Leu Gln Leu Ser Lys Asp Thr Tyr Asp
260 265 270
Asp Asp Leu Asp Asn Leu Leu Ala Gln Ile Gly Asp Gln Tyr Ala Asp
275 280 285
Leu Phe Leu Ala Ala Lys Asn Leu Ser Asp Ala Ile Leu Leu Ser Asp
290 295 300
Ile Leu Arg Val Asn Thr Glu Ile Thr Lys Ala Pro Leu Ser Ala Ser
305 310 315 320
Met Ile Lys Arg Tyr Asp Glu His His Gln Asp Leu Thr Leu Leu Lys
325 330 335
Ala Leu Val Arg Gln Gln Leu Pro Glu Lys Tyr Lys Glu Ile Phe Phe
340 345 350
Asp Gln Ser Lys Asn Gly Tyr Ala Gly Tyr Ile Asp Gly Gly Ala Ser
355 360 365
Gln Glu Glu Phe Tyr Lys Phe Ile Lys Pro Ile Leu Glu Lys Met Asp
370 375 380
Gly Thr Glu Glu Leu Leu Val Lys Leu Asn Arg Glu Asp Leu Leu Arg
385 390 395 400
Lys Gln Arg Thr Phe Asp Asn Gly Ser Ile Pro His Gln Ile His Leu
405 410 415
Gly Glu Leu His Ala Ile Leu Arg Arg Gln Glu Asp Phe Tyr Pro Phe
420 425 430
Leu Lys Asp Asn Arg Glu Lys Ile Glu Lys Ile Leu Thr Phe Arg Ile
435 440 445
Pro Tyr Tyr Val Gly Pro Leu Ala Arg Gly Asn Ser Arg Phe Ala Trp
450 455 460
Met Thr Arg Lys Ser Glu Glu Thr Ile Thr Pro Trp Asn Phe Glu Glu
465 470 475 480
Val Val Asp Lys Gly Ala Ser Ala Gln Ser Phe Ile Glu Arg Met Thr
485 490 495
Asn Phe Asp Lys Asn Leu Pro Asn Glu Lys Val Leu Pro Lys His Ser
500 505 510
Leu Leu Tyr Glu Tyr Phe Thr Val Tyr Asn Glu Leu Thr Lys Val Lys
515 520 525
Tyr Val Thr Glu Gly Met Arg Lys Pro Ala Phe Leu Ser Gly Glu Gln
530 535 540
Lys Lys Ala Ile Val Asp Leu Leu Phe Lys Thr Asn Arg Lys Val Thr
545 550 555 560
Val Lys Gln Leu Lys Glu Asp Tyr Phe Lys Lys Ile Glu Cys Phe Asp
565 570 575
Ser Val Glu Ile Ser Gly Val Glu Asp Arg Phe Asn Ala Ser Leu Gly
580 585 590
Thr Tyr His Asp Leu Leu Lys Ile Ile Lys Asp Lys Asp Phe Leu Asp
595 600 605
Asn Glu Glu Asn Glu Asp Ile Leu Glu Asp Ile Val Leu Thr Leu Thr
610 615 620
Leu Phe Glu Asp Arg Glu Met Ile Glu Glu Arg Leu Lys Thr Tyr Ala
625 630 635 640
His Leu Phe Asp Asp Lys Val Met Lys Gln Leu Lys Arg Arg Arg Tyr
645 650 655
Thr Gly Trp Gly Arg Leu Ser Arg Lys Leu Ile Asn Gly Ile Arg Asp
660 665 670
Lys Gln Ser Gly Lys Thr Ile Leu Asp Phe Leu Lys Ser Asp Gly Phe
675 680 685
Ala Asn Arg Asn Phe Met Gln Leu Ile His Asp Asp Ser Leu Thr Phe
690 695 700
Lys Glu Asp Ile Gln Lys Ala Gln Val Ser Gly Gln Gly Asp Ser Leu
705 710 715 720
His Glu His Ile Ala Asn Leu Ala Gly Ser Pro Ala Ile Lys Lys Gly
725 730 735
Ile Leu Gln Thr Val Lys Val Val Asp Glu Leu Val Lys Val Met Gly
740 745 750
Arg His Lys Pro Glu Asn Ile Val Ile Glu Met Ala Arg Glu Asn Gln
755 760 765
Thr Thr Gln Lys Gly Gln Lys Asn Ser Arg Glu Arg Met Lys Arg Ile
770 775 780
Glu Glu Gly Ile Lys Glu Leu Gly Ser Gln Ile Leu Lys Glu His Pro
785 790 795 800
Val Glu Asn Thr Gln Leu Gln Asn Glu Lys Leu Tyr Leu Tyr Tyr Leu
805 810 815
Gln Asn Gly Arg Asp Met Tyr Val Asp Gln Glu Leu Asp Ile Asn Arg
820 825 830
Leu Ser Asp Tyr Asp Val Asp Ala Ile Val Pro Gln Ser Phe Leu Lys
835 840 845
Asp Asp Ser Ile Asp Asn Lys Val Leu Thr Arg Ser Asp Lys Asn Arg
850 855 860
Gly Lys Ser Asp Asn Val Pro Ser Glu Glu Val Val Lys Lys Met Lys
865 870 875 880
Asn Tyr Trp Arg Gln Leu Leu Asn Ala Lys Leu Ile Thr Gln Arg Lys
885 890 895
Phe Asp Asn Leu Thr Lys Ala Glu Arg Gly Gly Leu Ser Glu Leu Asp
900 905 910
Lys Ala Gly Phe Ile Lys Arg Gln Leu Val Glu Thr Arg Gln Ile Thr
915 920 925
Lys His Val Ala Gln Ile Leu Asp Ser Arg Met Asn Thr Lys Tyr Asp
930 935 940
Glu Asn Asp Lys Leu Ile Arg Glu Val Lys Val Ile Thr Leu Lys Ser
945 950 955 960
Lys Leu Val Ser Asp Phe Arg Lys Asp Phe Gln Phe Tyr Lys Val Arg
965 970 975
Glu Ile Asn Asn Tyr His His Ala His Asp Ala Tyr Leu Asn Ala Val
980 985 990
Val Gly Thr Ala Leu Ile Lys Lys Tyr Pro Lys Leu Glu Ser Glu Phe
995 1000 1005
Val Tyr Gly Asp Tyr Lys Val Tyr Asp Val Arg Lys Met Ile Ala
1010 1015 1020
Lys Ser Glu Gln Glu Ile Gly Lys Ala Thr Ala Lys Tyr Phe Phe
1025 1030 1035
Tyr Ser Asn Ile Met Asn Phe Phe Lys Thr Glu Ile Thr Leu Ala
1040 1045 1050
Asn Gly Glu Ile Arg Lys Arg Pro Leu Ile Glu Thr Asn Gly Glu
1055 1060 1065
Thr Gly Glu Ile Val Trp Asp Lys Gly Arg Asp Phe Ala Thr Val
1070 1075 1080
Arg Lys Val Leu Ser Met Pro Gln Val Asn Ile Val Lys Lys Thr
1085 1090 1095
Glu Val Gln Thr Gly Gly Phe Ser Lys Glu Ser Ile Leu Pro Lys
1100 1105 1110
Arg Asn Ser Asp Lys Leu Ile Ala Arg Lys Lys Asp Trp Asp Pro
1115 1120 1125
Lys Lys Tyr Gly Gly Phe Asp Ser Pro Thr Val Ala Tyr Ser Val
1130 1135 1140
Leu Val Val Ala Lys Val Glu Lys Gly Lys Ser Lys Lys Leu Lys
1145 1150 1155
Ser Val Lys Glu Leu Leu Gly Ile Thr Ile Met Glu Arg Ser Ser
1160 1165 1170
Phe Glu Lys Asn Pro Ile Asp Phe Leu Glu Ala Lys Gly Tyr Lys
1175 1180 1185
Glu Val Lys Lys Asp Leu Ile Ile Lys Leu Pro Lys Tyr Ser Leu
1190 1195 1200
Phe Glu Leu Glu Asn Gly Arg Lys Arg Met Leu Ala Ser Ala Gly
1205 1210 1215
Glu Leu Gln Lys Gly Asn Glu Leu Ala Leu Pro Ser Lys Tyr Val
1220 1225 1230
Asn Phe Leu Tyr Leu Ala Ser His Tyr Glu Lys Leu Lys Gly Ser
1235 1240 1245
Pro Glu Asp Asn Glu Gln Lys Gln Leu Phe Val Glu Gln His Lys
1250 1255 1260
His Tyr Leu Asp Glu Ile Ile Glu Gln Ile Ser Glu Phe Ser Lys
1265 1270 1275
Arg Val Ile Leu Ala Asp Ala Asn Leu Asp Lys Val Leu Ser Ala
1280 1285 1290
Tyr Asn Lys His Arg Asp Lys Pro Ile Arg Glu Gln Ala Glu Asn
1295 1300 1305
Ile Ile His Leu Phe Thr Leu Thr Asn Leu Gly Ala Pro Ala Ala
1310 1315 1320
Phe Lys Tyr Phe Asp Thr Thr Ile Asp Arg Lys Arg Tyr Thr Ser
1325 1330 1335
Thr Lys Glu Val Leu Asp Ala Thr Leu Ile His Gln Ser Ile Thr
1340 1345 1350
Gly Leu Tyr Glu Thr Arg Ile Asp Leu Ser Gln Leu Gly Gly Asp
1355 1360 1365
<210> 24
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетический полипептид
<400> 24
Tyr Pro Tyr Asp Val Pro Asp Tyr Ala
1. 5
<210> 25
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетический полипептид
<400> 25
Pro Lys Lys Lys Arg Lys Val
1. 5
<210> 26
<211> 301
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетический полипептид
<400> 26
Asn His Asp Gln Glu Phe Asp Pro Pro Lys Val Tyr Pro Pro Val Pro
1. 5 10 15
Ala Glu Lys Arg Lys Pro Ile Arg Val Leu Ser Leu Phe Asp Gly Ile
20 25 30
Ala Thr Gly Leu Leu Val Leu Lys Asp Leu Gly Ile Gln Val Asp Arg
35 40 45
Tyr Ile Ala Ser Glu Val Cys Glu Asp Ser Ile Thr Val Gly Met Val
50 55 60
Arg His Gln Gly Lys Ile Met Tyr Val Gly Asp Val Arg Ser Val Thr
65 70 75 80
Gln Lys His Ile Gln Glu Trp Gly Pro Phe Asp Leu Val Ile Gly Gly
85 90 95
Ser Pro Cys Asn Asp Leu Ser Ile Val Asn Pro Ala Arg Lys Gly Leu
100 105 110
Tyr Glu Gly Thr Gly Arg Leu Phe Phe Glu Phe Tyr Arg Leu Leu His
115 120 125
Asp Ala Arg Pro Lys Glu Gly Asp Asp Arg Pro Phe Phe Trp Leu Phe
130 135 140
Glu Asn Val Val Ala Met Gly Val Ser Asp Lys Arg Asp Ile Ser Arg
145 150 155 160
Phe Leu Glu Ser Asn Pro Val Met Ile Asp Ala Lys Glu Val Ser Ala
165 170 175
Ala His Arg Ala Arg Tyr Phe Trp Gly Asn Leu Pro Gly Met Asn Arg
180 185 190
Pro Leu Ala Ser Thr Val Asn Asp Lys Leu Glu Leu Gln Glu Cys Leu
195 200 205
Glu His Gly Arg Ile Ala Lys Phe Ser Lys Val Arg Thr Ile Thr Thr
210 215 220
Arg Ser Asn Ser Ile Lys Gln Gly Lys Asp Gln His Phe Pro Val Phe
225 230 235 240
Met Asn Glu Lys Glu Asp Ile Leu Trp Cys Thr Glu Met Glu Arg Val
245 250 255
Phe Gly Phe Pro Val His Tyr Thr Asp Val Ser Asn Met Ser Arg Leu
260 265 270
Ala Arg Gln Arg Leu Leu Gly Arg Ser Trp Ser Val Pro Val Ile Arg
275 280 285
His Leu Phe Ala Pro Leu Lys Glu Tyr Phe Ala Cys Val
290 295 300
<210> 27
<211> 27
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетический полипептид
<400> 27
Ser Ser Gly Asn Ser Asn Ala Asn Ser Arg Gly Pro Ser Phe Ser Ser
1. 5 10 15
Gly Leu Val Pro Leu Ser Leu Arg Gly Ser His
20 25
<210> 28
<211> 215
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетический полипептид
<400> 28
Met Gly Pro Met Glu Ile Tyr Lys Thr Val Ser Ala Trp Lys Arg Gln
1. 5 10 15
Pro Val Arg Val Leu Ser Leu Phe Arg Asn Ile Asp Lys Val Leu Lys
20 25 30
Ser Leu Gly Phe Leu Glu Ser Gly Ser Gly Ser Gly Gly Gly Thr Leu
35 40 45
Lys Tyr Val Glu Asp Val Thr Asn Val Val Arg Arg Asp Val Glu Lys
50 55 60
Trp Gly Pro Phe Asp Leu Val Tyr Gly Ser Thr Gln Pro Leu Gly Ser
65 70 75 80
Ser Cys Asp Arg Cys Pro Gly Trp Tyr Met Phe Gln Phe His Arg Ile
85 90 95
Leu Gln Tyr Ala Leu Pro Arg Gln Glu Ser Gln Arg Pro Phe Phe Trp
100 105 110
Ile Phe Met Asp Asn Leu Leu Leu Thr Glu Asp Asp Gln Glu Thr Thr
115 120 125
Thr Arg Phe Leu Gln Thr Glu Ala Val Thr Leu Gln Asp Val Arg Gly
130 135 140
Arg Asp Tyr Gln Asn Ala Met Arg Val Trp Ser Asn Ile Pro Gly Leu
145 150 155 160
Lys Ser Lys His Ala Pro Leu Thr Pro Lys Glu Glu Glu Tyr Leu Gln
165 170 175
Ala Gln Val Arg Ser Arg Ser Lys Leu Asp Ala Pro Lys Val Asp Leu
180 185 190
Leu Val Lys Asn Cys Leu Leu Pro Leu Arg Glu Tyr Phe Lys Tyr Phe
195 200 205
Ser Gln Asn Ser Leu Pro Leu
210 215
<210> 29
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетический полипептид
<400> 29
Ala Thr Asn Phe Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn
1. 5 10 15
Pro Gly Pro
<210> 30
<211> 232
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетический полипептид
<400> 30
Ser Glu Leu Ile Lys Glu Asn Met His Met Lys Leu Tyr Met Glu Gly
1. 5 10 15
Thr Val Asp Asn His His Phe Lys Cys Thr Ser Glu Gly Glu Gly Lys
20 25 30
Pro Tyr Glu Gly Thr Gln Thr Met Arg Ile Lys Val Val Glu Gly Gly
35 40 45
Pro Leu Pro Phe Ala Phe Asp Ile Leu Ala Thr Ser Phe Leu Tyr Gly
50 55 60
Ser Lys Thr Phe Ile Asn His Thr Gln Gly Ile Pro Asp Phe Phe Lys
65 70 75 80
Gln Ser Phe Pro Glu Gly Phe Thr Trp Glu Arg Val Thr Thr Tyr Glu
85 90 95
Asp Gly Gly Val Leu Thr Ala Thr Gln Asp Thr Ser Leu Gln Asp Gly
100 105 110
Cys Leu Ile Tyr Asn Val Lys Ile Arg Gly Val Asn Phe Thr Ser Asn
115 120 125
Gly Pro Val Met Gln Lys Lys Thr Leu Gly Trp Glu Ala Phe Thr Glu
130 135 140
Thr Leu Tyr Pro Ala Asp Gly Gly Leu Glu Gly Arg Asn Asp Met Ala
145 150 155 160
Leu Lys Leu Val Gly Gly Ser His Leu Ile Ala Asn Ile Lys Thr Thr
165 170 175
Tyr Arg Ser Lys Lys Pro Ala Lys Asn Leu Lys Met Pro Gly Val Tyr
180 185 190
Tyr Val Asp Tyr Arg Leu Glu Arg Ile Lys Glu Ala Asn Asn Glu Thr
195 200 205
Tyr Val Glu Gln His Glu Val Ala Val Ala Arg Tyr Cys Asp Leu Pro
210 215 220
Ser Lys Leu Gly His Lys Leu Asn
225 230
<210> 31
<211> 16
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетический полипептид
<400> 31
Ser Gly Ser Glu Thr Pro Gly Thr Ser Glu Ser Ala Thr Pro Glu Ser
1. 5 10 15
<210> 32
<211> 80
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетический полипептид
<400> 32
Gly Gly Pro Ser Ser Gly Ala Pro Pro Pro Ser Gly Gly Ser Pro Ala
1. 5 10 15
Gly Ser Pro Thr Ser Thr Glu Glu Gly Thr Ser Glu Ser Ala Thr Pro
20 25 30
Glu Ser Gly Pro Gly Thr Ser Thr Glu Pro Ser Glu Gly Ser Ala Pro
35 40 45
Gly Ser Pro Ala Gly Ser Pro Thr Ser Thr Glu Glu Gly Thr Ser Thr
50 55 60
Glu Pro Ser Glu Gly Ser Ala Pro Gly Thr Ser Thr Glu Pro Ser Glu
65 70 75 80
<210> 33
<211> 543
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетический полипептид
<400> 33
Asn His Asp Gln Glu Phe Asp Pro Pro Lys Val Tyr Pro Pro Val Pro
1. 5 10 15
Ala Glu Lys Arg Lys Pro Ile Arg Val Leu Ser Leu Phe Asp Gly Ile
20 25 30
Ala Thr Gly Leu Leu Val Leu Lys Asp Leu Gly Ile Gln Val Asp Arg
35 40 45
Tyr Ile Ala Ser Glu Val Cys Glu Asp Ser Ile Thr Val Gly Met Val
50 55 60
Arg His Gln Gly Lys Ile Met Tyr Val Gly Asp Val Arg Ser Val Thr
65 70 75 80
Gln Lys His Ile Gln Glu Trp Gly Pro Phe Asp Leu Val Ile Gly Gly
85 90 95
Ser Pro Cys Asn Asp Leu Ser Ile Val Asn Pro Ala Arg Lys Gly Leu
100 105 110
Tyr Glu Gly Thr Gly Arg Leu Phe Phe Glu Phe Tyr Arg Leu Leu His
115 120 125
Asp Ala Arg Pro Lys Glu Gly Asp Asp Arg Pro Phe Phe Trp Leu Phe
130 135 140
Glu Asn Val Val Ala Met Gly Val Ser Asp Lys Arg Asp Ile Ser Arg
145 150 155 160
Phe Leu Glu Ser Asn Pro Val Met Ile Asp Ala Lys Glu Val Ser Ala
165 170 175
Ala His Arg Ala Arg Tyr Phe Trp Gly Asn Leu Pro Gly Met Asn Arg
180 185 190
Pro Leu Ala Ser Thr Val Asn Asp Lys Leu Glu Leu Gln Glu Cys Leu
195 200 205
Glu His Gly Arg Ile Ala Lys Phe Ser Lys Val Arg Thr Ile Thr Thr
210 215 220
Arg Ser Asn Ser Ile Lys Gln Gly Lys Asp Gln His Phe Pro Val Phe
225 230 235 240
Met Asn Glu Lys Glu Asp Ile Leu Trp Cys Thr Glu Met Glu Arg Val
245 250 255
Phe Gly Phe Pro Val His Tyr Thr Asp Val Ser Asn Met Ser Arg Leu
260 265 270
Ala Arg Gln Arg Leu Leu Gly Arg Ser Trp Ser Val Pro Val Ile Arg
275 280 285
His Leu Phe Ala Pro Leu Lys Glu Tyr Phe Ala Cys Val Ser Ser Gly
290 295 300
Asn Ser Asn Ala Asn Ser Arg Gly Pro Ser Phe Ser Ser Gly Leu Val
305 310 315 320
Pro Leu Ser Leu Arg Gly Ser His Met Gly Pro Met Glu Ile Tyr Lys
325 330 335
Thr Val Ser Ala Trp Lys Arg Gln Pro Val Arg Val Leu Ser Leu Phe
340 345 350
Arg Asn Ile Asp Lys Val Leu Lys Ser Leu Gly Phe Leu Glu Ser Gly
355 360 365
Ser Gly Ser Gly Gly Gly Thr Leu Lys Tyr Val Glu Asp Val Thr Asn
370 375 380
Val Val Arg Arg Asp Val Glu Lys Trp Gly Pro Phe Asp Leu Val Tyr
385 390 395 400
Gly Ser Thr Gln Pro Leu Gly Ser Ser Cys Asp Arg Cys Pro Gly Trp
405 410 415
Tyr Met Phe Gln Phe His Arg Ile Leu Gln Tyr Ala Leu Pro Arg Gln
420 425 430
Glu Ser Gln Arg Pro Phe Phe Trp Ile Phe Met Asp Asn Leu Leu Leu
435 440 445
Thr Glu Asp Asp Gln Glu Thr Thr Thr Arg Phe Leu Gln Thr Glu Ala
450 455 460
Val Thr Leu Gln Asp Val Arg Gly Arg Asp Tyr Gln Asn Ala Met Arg
465 470 475 480
Val Trp Ser Asn Ile Pro Gly Leu Lys Ser Lys His Ala Pro Leu Thr
485 490 495
Pro Lys Glu Glu Glu Tyr Leu Gln Ala Gln Val Arg Ser Arg Ser Lys
500 505 510
Leu Asp Ala Pro Lys Val Asp Leu Leu Val Lys Asn Cys Leu Leu Pro
515 520 525
Leu Arg Glu Tyr Phe Lys Tyr Phe Ser Gln Asn Ser Leu Pro Leu
530 535 540
<210> 34
<211> 1307
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетический полипептид
<400> 34
Met Thr Gln Phe Glu Gly Phe Thr Asn Leu Tyr Gln Val Ser Lys Thr
1. 5 10 15
Leu Arg Phe Glu Leu Ile Pro Gln Gly Lys Thr Leu Lys His Ile Gln
20 25 30
Glu Gln Gly Phe Ile Glu Glu Asp Lys Ala Arg Asn Asp His Tyr Lys
35 40 45
Glu Leu Lys Pro Ile Ile Asp Arg Ile Tyr Lys Thr Tyr Ala Asp Gln
50 55 60
Cys Leu Gln Leu Val Gln Leu Asp Trp Glu Asn Leu Ser Ala Ala Ile
65 70 75 80
Asp Ser Tyr Arg Lys Glu Lys Thr Glu Glu Thr Arg Asn Ala Leu Ile
85 90 95
Glu Glu Gln Ala Thr Tyr Arg Asn Ala Ile His Asp Tyr Phe Ile Gly
100 105 110
Arg Thr Asp Asn Leu Thr Asp Ala Ile Asn Lys Arg His Ala Glu Ile
115 120 125
Tyr Lys Gly Leu Phe Lys Ala Glu Leu Phe Asn Gly Lys Val Leu Lys
130 135 140
Gln Leu Gly Thr Val Thr Thr Thr Glu His Glu Asn Ala Leu Leu Arg
145 150 155 160
Ser Phe Asp Lys Phe Thr Thr Tyr Phe Ser Gly Phe Tyr Glu Asn Arg
165 170 175
Lys Asn Val Phe Ser Ala Glu Asp Ile Ser Thr Ala Ile Pro His Arg
180 185 190
Ile Val Gln Asp Asn Phe Pro Lys Phe Lys Glu Asn Cys His Ile Phe
195 200 205
Thr Arg Leu Ile Thr Ala Val Pro Ser Leu Arg Glu His Phe Glu Asn
210 215 220
Val Lys Lys Ala Ile Gly Ile Phe Val Ser Thr Ser Ile Glu Glu Val
225 230 235 240
Phe Ser Phe Pro Phe Tyr Asn Gln Leu Leu Thr Gln Thr Gln Ile Asp
245 250 255
Leu Tyr Asn Gln Leu Leu Gly Gly Ile Ser Arg Glu Ala Gly Thr Glu
260 265 270
Lys Ile Lys Gly Leu Asn Glu Val Leu Asn Leu Ala Ile Gln Lys Asn
275 280 285
Asp Glu Thr Ala His Ile Ile Ala Ser Leu Pro His Arg Phe Ile Pro
290 295 300
Leu Phe Lys Gln Ile Leu Ser Asp Arg Asn Thr Leu Ser Phe Ile Leu
305 310 315 320
Glu Glu Phe Lys Ser Asp Glu Glu Val Ile Gln Ser Phe Cys Lys Tyr
325 330 335
Lys Thr Leu Leu Arg Asn Glu Asn Val Leu Glu Thr Ala Glu Ala Leu
340 345 350
Phe Asn Glu Leu Asn Ser Ile Asp Leu Thr His Ile Phe Ile Ser His
355 360 365
Lys Lys Leu Glu Thr Ile Ser Ser Ala Leu Cys Asp His Trp Asp Thr
370 375 380
Leu Arg Asn Ala Leu Tyr Glu Arg Arg Ile Ser Glu Leu Thr Gly Lys
385 390 395 400
Ile Thr Lys Ser Ala Lys Glu Lys Val Gln Arg Ser Leu Lys His Glu
405 410 415
Asp Ile Asn Leu Gln Glu Ile Ile Ser Ala Ala Gly Lys Glu Leu Ser
420 425 430
Glu Ala Phe Lys Gln Lys Thr Ser Glu Ile Leu Ser His Ala His Ala
435 440 445
Ala Leu Asp Gln Pro Leu Pro Thr Thr Leu Lys Lys Gln Glu Glu Lys
450 455 460
Glu Ile Leu Lys Ser Gln Leu Asp Ser Leu Leu Gly Leu Tyr His Leu
465 470 475 480
Leu Asp Trp Phe Ala Val Asp Glu Ser Asn Glu Val Asp Pro Glu Phe
485 490 495
Ser Ala Arg Leu Thr Gly Ile Lys Leu Glu Met Glu Pro Ser Leu Ser
500 505 510
Phe Tyr Asn Lys Ala Arg Asn Tyr Ala Thr Lys Lys Pro Tyr Ser Val
515 520 525
Glu Lys Phe Lys Leu Asn Phe Gln Met Pro Thr Leu Ala Ser Gly Trp
530 535 540
Asp Val Asn Lys Glu Lys Asn Asn Gly Ala Ile Leu Phe Val Lys Asn
545 550 555 560
Gly Leu Tyr Tyr Leu Gly Ile Met Pro Lys Gln Lys Gly Arg Tyr Lys
565 570 575
Ala Leu Ser Phe Glu Pro Thr Glu Lys Thr Ser Glu Gly Phe Asp Lys
580 585 590
Met Tyr Tyr Asp Tyr Phe Pro Asp Ala Ala Lys Met Ile Pro Lys Cys
595 600 605
Ser Thr Gln Leu Lys Ala Val Thr Ala His Phe Gln Thr His Thr Thr
610 615 620
Pro Ile Leu Leu Ser Asn Asn Phe Ile Glu Pro Leu Glu Ile Thr Lys
625 630 635 640
Glu Ile Tyr Asp Leu Asn Asn Pro Glu Lys Glu Pro Lys Lys Phe Gln
645 650 655
Thr Ala Tyr Ala Lys Lys Thr Gly Asp Gln Lys Gly Tyr Arg Glu Ala
660 665 670
Leu Cys Lys Trp Ile Asp Phe Thr Arg Asp Phe Leu Ser Lys Tyr Thr
675 680 685
Lys Thr Thr Ser Ile Asp Leu Ser Ser Leu Arg Pro Ser Ser Gln Tyr
690 695 700
Lys Asp Leu Gly Glu Tyr Tyr Ala Glu Leu Asn Pro Leu Leu Tyr His
705 710 715 720
Ile Ser Phe Gln Arg Ile Ala Glu Lys Glu Ile Met Asp Ala Val Glu
725 730 735
Thr Gly Lys Leu Tyr Leu Phe Gln Ile Tyr Asn Lys Asp Phe Ala Lys
740 745 750
Gly His His Gly Lys Pro Asn Leu His Thr Leu Tyr Trp Thr Gly Leu
755 760 765
Phe Ser Pro Glu Asn Leu Ala Lys Thr Ser Ile Lys Leu Asn Gly Gln
770 775 780
Ala Glu Leu Phe Tyr Arg Pro Lys Ser Arg Met Lys Arg Met Ala His
785 790 795 800
Arg Leu Gly Glu Lys Met Leu Asn Lys Lys Leu Lys Asp Gln Lys Thr
805 810 815
Pro Ile Pro Asp Thr Leu Tyr Gln Glu Leu Tyr Asp Tyr Val Asn His
820 825 830
Arg Leu Ser His Asp Leu Ser Asp Glu Ala Arg Ala Leu Leu Pro Asn
835 840 845
Val Ile Thr Lys Glu Val Ser His Glu Ile Ile Lys Asp Arg Arg Phe
850 855 860
Thr Ser Asp Lys Phe Phe Phe His Val Pro Ile Thr Leu Asn Tyr Gln
865 870 875 880
Ala Ala Asn Ser Pro Ser Lys Phe Asn Gln Arg Val Asn Ala Tyr Leu
885 890 895
Lys Glu His Pro Glu Thr Pro Ile Ile Gly Ile Asp Arg Gly Glu Arg
900 905 910
Asn Leu Ile Tyr Ile Thr Val Ile Asp Ser Thr Gly Lys Ile Leu Glu
915 920 925
Gln Arg Ser Leu Asn Thr Ile Gln Gln Phe Asp Tyr Gln Lys Lys Leu
930 935 940
Asp Asn Arg Glu Lys Glu Arg Val Ala Ala Arg Gln Ala Trp Ser Val
945 950 955 960
Val Gly Thr Ile Lys Asp Leu Lys Gln Gly Tyr Leu Ser Gln Val Ile
965 970 975
His Glu Ile Val Asp Leu Met Ile His Tyr Gln Ala Val Val Val Leu
980 985 990
Ala Asn Leu Asn Phe Gly Phe Lys Ser Lys Arg Thr Gly Ile Ala Glu
995 1000 1005
Lys Ala Val Tyr Gln Gln Phe Glu Lys Met Leu Ile Asp Lys Leu
1010 1015 1020
Asn Cys Leu Val Leu Lys Asp Tyr Pro Ala Glu Lys Val Gly Gly
1025 1030 1035
Val Leu Asn Pro Tyr Gln Leu Thr Asp Gln Phe Thr Ser Phe Ala
1040 1045 1050
Lys Met Gly Thr Gln Ser Gly Phe Leu Phe Tyr Val Pro Ala Pro
1055 1060 1065
Tyr Thr Ser Lys Ile Asp Pro Leu Thr Gly Phe Val Asp Pro Phe
1070 1075 1080
Val Trp Lys Thr Ile Lys Asn His Glu Ser Arg Lys His Phe Leu
1085 1090 1095
Glu Gly Phe Asp Phe Leu His Tyr Asp Val Lys Thr Gly Asp Phe
1100 1105 1110
Ile Leu His Phe Lys Met Asn Arg Asn Leu Ser Phe Gln Arg Gly
1115 1120 1125
Leu Pro Gly Phe Met Pro Ala Trp Asp Ile Val Phe Glu Lys Asn
1130 1135 1140
Glu Thr Gln Phe Asp Ala Lys Gly Thr Pro Phe Ile Ala Gly Lys
1145 1150 1155
Arg Ile Val Pro Val Ile Glu Asn His Arg Phe Thr Gly Arg Tyr
1160 1165 1170
Arg Asp Leu Tyr Pro Ala Asn Glu Leu Ile Ala Leu Leu Glu Glu
1175 1180 1185
Lys Gly Ile Val Phe Arg Asp Gly Ser Asn Ile Leu Pro Lys Leu
1190 1195 1200
Leu Glu Asn Asp Asp Ser His Ala Ile Asp Thr Met Val Ala Leu
1205 1210 1215
Ile Arg Ser Val Leu Gln Met Arg Asn Ser Asn Ala Ala Thr Gly
1220 1225 1230
Glu Asp Tyr Ile Asn Ser Pro Val Arg Asp Leu Asn Gly Val Cys
1235 1240 1245
Phe Asp Ser Arg Phe Gln Asn Pro Glu Trp Pro Met Asp Ala Asp
1250 1255 1260
Ala Asn Gly Ala Tyr His Ile Ala Leu Lys Gly Gln Leu Leu Leu
1265 1270 1275
Asn His Leu Lys Glu Ser Lys Asp Leu Lys Leu Gln Asn Gly Ile
1280 1285 1290
Ser Asn Gln Asp Trp Leu Ala Tyr Ile Gln Glu Leu Arg Asn
1295 1300 1305
<210> 35
<211> 1228
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетический полипептид
<400> 35
Met Ser Lys Leu Glu Lys Phe Thr Asn Cys Tyr Ser Leu Ser Lys Thr
1. 5 10 15
Leu Arg Phe Lys Ala Ile Pro Val Gly Lys Thr Gln Glu Asn Ile Asp
20 25 30
Asn Lys Arg Leu Leu Val Glu Asp Glu Lys Arg Ala Glu Asp Tyr Lys
35 40 45
Gly Val Lys Lys Leu Leu Asp Arg Tyr Tyr Leu Ser Phe Ile Asn Asp
50 55 60
Val Leu His Ser Ile Lys Leu Lys Asn Leu Asn Asn Tyr Ile Ser Leu
65 70 75 80
Phe Arg Lys Lys Thr Arg Thr Glu Lys Glu Asn Lys Glu Leu Glu Asn
85 90 95
Leu Glu Ile Asn Leu Arg Lys Glu Ile Ala Lys Ala Phe Lys Gly Asn
100 105 110
Glu Gly Tyr Lys Ser Leu Phe Lys Lys Asp Ile Ile Glu Thr Ile Leu
115 120 125
Pro Glu Phe Leu Asp Asp Lys Asp Glu Ile Ala Leu Val Asn Ser Phe
130 135 140
Asn Gly Phe Thr Thr Ala Phe Thr Gly Phe Phe Asp Asn Arg Glu Asn
145 150 155 160
Met Phe Ser Glu Glu Ala Lys Ser Thr Ser Ile Ala Phe Arg Cys Ile
165 170 175
Asn Glu Asn Leu Thr Arg Tyr Ile Ser Asn Met Asp Ile Phe Glu Lys
180 185 190
Val Asp Ala Ile Phe Asp Lys His Glu Val Gln Glu Ile Lys Glu Lys
195 200 205
Ile Leu Asn Ser Asp Tyr Asp Val Glu Asp Phe Phe Glu Gly Glu Phe
210 215 220
Phe Asn Phe Val Leu Thr Gln Glu Gly Ile Asp Val Tyr Asn Ala Ile
225 230 235 240
Ile Gly Gly Phe Val Thr Glu Ser Gly Glu Lys Ile Lys Gly Leu Asn
245 250 255
Glu Tyr Ile Asn Leu Tyr Asn Gln Lys Thr Lys Gln Lys Leu Pro Lys
260 265 270
Phe Lys Pro Leu Tyr Lys Gln Val Leu Ser Asp Arg Glu Ser Leu Ser
275 280 285
Phe Tyr Gly Glu Gly Tyr Thr Ser Asp Glu Glu Val Leu Glu Val Phe
290 295 300
Arg Asn Thr Leu Asn Lys Asn Ser Glu Ile Phe Ser Ser Ile Lys Lys
305 310 315 320
Leu Glu Lys Leu Phe Lys Asn Phe Asp Glu Tyr Ser Ser Ala Gly Ile
325 330 335
Phe Val Lys Asn Gly Pro Ala Ile Ser Thr Ile Ser Lys Asp Ile Phe
340 345 350
Gly Glu Trp Asn Val Ile Arg Asp Lys Trp Asn Ala Glu Tyr Asp Asp
355 360 365
Ile His Leu Lys Lys Lys Ala Val Val Thr Glu Lys Tyr Glu Asp Asp
370 375 380
Arg Arg Lys Ser Phe Lys Lys Ile Gly Ser Phe Ser Leu Glu Gln Leu
385 390 395 400
Gln Glu Tyr Ala Asp Ala Asp Leu Ser Val Val Glu Lys Leu Lys Glu
405 410 415
Ile Ile Ile Gln Lys Val Asp Glu Ile Tyr Lys Val Tyr Gly Ser Ser
420 425 430
Glu Lys Leu Phe Asp Ala Asp Phe Val Leu Glu Lys Ser Leu Lys Lys
435 440 445
Asn Asp Ala Val Val Ala Ile Met Lys Asp Leu Leu Asp Ser Val Lys
450 455 460
Ser Phe Glu Asn Tyr Ile Lys Ala Phe Phe Gly Glu Gly Lys Glu Thr
465 470 475 480
Asn Arg Asp Glu Ser Phe Tyr Gly Asp Phe Val Leu Ala Tyr Asp Ile
485 490 495
Leu Leu Lys Val Asp His Ile Tyr Asp Ala Ile Arg Asn Tyr Val Thr
500 505 510
Gln Lys Pro Tyr Ser Lys Asp Lys Phe Lys Leu Tyr Phe Gln Asn Pro
515 520 525
Gln Phe Met Gly Gly Trp Asp Lys Asp Lys Glu Thr Asp Tyr Arg Ala
530 535 540
Thr Ile Leu Arg Tyr Gly Ser Lys Tyr Tyr Leu Ala Ile Met Asp Lys
545 550 555 560
Lys Tyr Ala Lys Cys Leu Gln Lys Ile Asp Lys Asp Asp Val Asn Gly
565 570 575
Asn Tyr Glu Lys Ile Asn Tyr Lys Leu Leu Pro Gly Pro Asn Lys Met
580 585 590
Leu Pro Lys Val Phe Phe Ser Lys Lys Trp Met Ala Tyr Tyr Asn Pro
595 600 605
Ser Glu Asp Ile Gln Lys Ile Tyr Lys Asn Gly Thr Phe Lys Lys Gly
610 615 620
Asp Met Phe Asn Leu Asn Asp Cys His Lys Leu Ile Asp Phe Phe Lys
625 630 635 640
Asp Ser Ile Ser Arg Tyr Pro Lys Trp Ser Asn Ala Tyr Asp Phe Asn
645 650 655
Phe Ser Glu Thr Glu Lys Tyr Lys Asp Ile Ala Gly Phe Tyr Arg Glu
660 665 670
Val Glu Glu Gln Gly Tyr Lys Val Ser Phe Glu Ser Ala Ser Lys Lys
675 680 685
Glu Val Asp Lys Leu Val Glu Glu Gly Lys Leu Tyr Met Phe Gln Ile
690 695 700
Tyr Asn Lys Asp Phe Ser Asp Lys Ser His Gly Thr Pro Asn Leu His
705 710 715 720
Thr Met Tyr Phe Lys Leu Leu Phe Asp Glu Asn Asn His Gly Gln Ile
725 730 735
Arg Leu Ser Gly Gly Ala Glu Leu Phe Met Arg Arg Ala Ser Leu Lys
740 745 750
Lys Glu Glu Leu Val Val His Pro Ala Asn Ser Pro Ile Ala Asn Lys
755 760 765
Asn Pro Asp Asn Pro Lys Lys Thr Thr Thr Leu Ser Tyr Asp Val Tyr
770 775 780
Lys Asp Lys Arg Phe Ser Glu Asp Gln Tyr Glu Leu His Ile Pro Ile
785 790 795 800
Ala Ile Asn Lys Cys Pro Lys Asn Ile Phe Lys Ile Asn Thr Glu Val
805 810 815
Arg Val Leu Leu Lys His Asp Asp Asn Pro Tyr Val Ile Gly Ile Ala
820 825 830
Arg Gly Glu Arg Asn Leu Leu Tyr Ile Val Val Val Asp Gly Lys Gly
835 840 845
Asn Ile Val Glu Gln Tyr Ser Leu Asn Glu Ile Ile Asn Asn Phe Asn
850 855 860
Gly Ile Arg Ile Lys Thr Asp Tyr His Ser Leu Leu Asp Lys Lys Glu
865 870 875 880
Lys Glu Arg Phe Glu Ala Arg Gln Asn Trp Thr Ser Ile Glu Asn Ile
885 890 895
Lys Glu Leu Lys Ala Gly Tyr Ile Ser Gln Val Val His Lys Ile Cys
900 905 910
Glu Leu Val Glu Lys Tyr Asp Ala Val Ile Ala Leu Ala Asp Leu Asn
915 920 925
Ser Gly Phe Lys Asn Ser Arg Val Lys Val Glu Lys Gln Val Tyr Gln
930 935 940
Lys Phe Glu Lys Met Leu Ile Asp Lys Leu Asn Tyr Met Val Asp Lys
945 950 955 960
Lys Ser Asn Pro Cys Ala Thr Gly Gly Ala Leu Lys Gly Tyr Gln Ile
965 970 975
Thr Asn Lys Phe Glu Ser Phe Lys Ser Met Ser Thr Gln Asn Gly Phe
980 985 990
Ile Phe Tyr Ile Pro Ala Trp Leu Thr Ser Lys Ile Asp Pro Ser Thr
995 1000 1005
Gly Phe Val Asn Leu Leu Lys Thr Lys Tyr Thr Ser Ile Ala Asp
1010 1015 1020
Ser Lys Lys Phe Ile Ser Ser Phe Asp Arg Ile Met Tyr Val Pro
1025 1030 1035
Glu Glu Asp Leu Phe Glu Phe Ala Leu Asp Tyr Lys Asn Phe Ser
1040 1045 1050
Arg Thr Asp Ala Asp Tyr Ile Lys Lys Trp Lys Leu Tyr Ser Tyr
1055 1060 1065
Gly Asn Arg Ile Arg Ile Phe Arg Asn Pro Lys Lys Asn Asn Val
1070 1075 1080
Phe Asp Trp Glu Glu Val Cys Leu Thr Ser Ala Tyr Lys Glu Leu
1085 1090 1095
Phe Asn Lys Tyr Gly Ile Asn Tyr Gln Gln Gly Asp Ile Arg Ala
1100 1105 1110
Leu Leu Cys Glu Gln Ser Asp Lys Ala Phe Tyr Ser Ser Phe Met
1115 1120 1125
Ala Leu Met Ser Leu Met Leu Gln Met Arg Asn Ser Ile Thr Gly
1130 1135 1140
Arg Thr Asp Val Asp Phe Leu Ile Ser Pro Val Lys Asn Ser Asp
1145 1150 1155
Gly Ile Phe Tyr Asp Ser Arg Asn Tyr Glu Ala Gln Glu Asn Ala
1160 1165 1170
Ile Leu Pro Lys Asn Ala Asp Ala Asn Gly Ala Tyr Asn Ile Ala
1175 1180 1185
Arg Lys Val Leu Trp Ala Ile Gly Gln Phe Lys Lys Ala Glu Asp
1190 1195 1200
Glu Lys Leu Asp Lys Val Lys Ile Ala Ile Ser Asn Lys Glu Trp
1205 1210 1215
Leu Glu Tyr Ala Gln Thr Ser Val Lys His
1220 1225
<210> 36
<211> 1314
<212> Белок
<213> Искусственная последовательность
<220>
<223> Синтетический полипептид
<400> 36
Met Tyr Pro Tyr Asp Val Pro Asp Tyr Ala Ser Gly Ser Gly Met Ser
1. 5 10 15
Ile Tyr Gln Glu Phe Val Asn Lys Tyr Ser Leu Ser Lys Thr Leu Arg
20 25 30
Phe Glu Leu Ile Pro Gln Gly Lys Thr Leu Glu Asn Ile Lys Ala Arg
35 40 45
Gly Leu Ile Leu Asp Asp Glu Lys Arg Ala Lys Asp Tyr Lys Lys Ala
50 55 60
Lys Gln Ile Ile Asp Lys Tyr His Gln Phe Phe Ile Glu Glu Ile Leu
65 70 75 80
Ser Ser Val Cys Ile Ser Glu Asp Leu Leu Gln Asn Tyr Ser Asp Val
85 90 95
Tyr Phe Lys Leu Lys Lys Ser Asp Asp Asp Asn Leu Gln Lys Asp Phe
100 105 110
Lys Ser Ala Lys Asp Thr Ile Lys Lys Gln Ile Ser Glu Tyr Ile Lys
115 120 125
Asp Ser Glu Lys Phe Lys Asn Leu Phe Asn Gln Asn Leu Ile Asp Ala
130 135 140
Lys Lys Gly Gln Glu Ser Asp Leu Ile Leu Trp Leu Lys Gln Ser Lys
145 150 155 160
Asp Asn Gly Ile Glu Leu Phe Lys Ala Asn Ser Asp Ile Thr Asp Ile
165 170 175
Asp Glu Ala Leu Glu Ile Ile Lys Ser Phe Lys Gly Trp Thr Thr Tyr
180 185 190
Phe Lys Gly Phe His Glu Asn Arg Lys Asn Val Tyr Ser Ser Asn Asp
195 200 205
Ile Pro Thr Ser Ile Ile Tyr Arg Ile Val Asp Asp Asn Leu Pro Lys
210 215 220
Phe Leu Glu Asn Lys Ala Lys Tyr Glu Ser Leu Lys Asp Lys Ala Pro
225 230 235 240
Glu Ala Ile Asn Tyr Glu Gln Ile Lys Lys Asp Leu Ala Glu Glu Leu
245 250 255
Thr Phe Asp Ile Asp Tyr Lys Thr Ser Glu Val Asn Gln Arg Val Phe
260 265 270
Ser Leu Asp Glu Val Phe Glu Ile Ala Asn Phe Asn Asn Tyr Leu Asn
275 280 285
Gln Ser Gly Ile Thr Lys Phe Asn Thr Ile Ile Gly Gly Lys Phe Val
290 295 300
Asn Gly Glu Asn Thr Lys Arg Lys Gly Ile Asn Glu Tyr Ile Asn Leu
305 310 315 320
Tyr Ser Gln Gln Ile Asn Asp Lys Thr Leu Lys Lys Tyr Lys Met Ser
325 330 335
Val Leu Phe Lys Gln Ile Leu Ser Asp Thr Glu Ser Lys Ser Phe Val
340 345 350
Ile Asp Lys Leu Glu Asp Asp Ser Asp Val Val Thr Thr Met Gln Ser
355 360 365
Phe Tyr Glu Gln Ile Ala Ala Phe Lys Thr Val Glu Glu Lys Ser Ile
370 375 380
Lys Glu Thr Leu Ser Leu Leu Phe Asp Asp Leu Lys Ala Gln Lys Leu
385 390 395 400
Asp Leu Ser Lys Ile Tyr Phe Lys Asn Asp Lys Ser Leu Thr Asp Leu
405 410 415
Ser Gln Gln Val Phe Asp Asp Tyr Ser Val Ile Gly Thr Ala Val Leu
420 425 430
Glu Tyr Ile Thr Gln Gln Ile Ala Pro Lys Asn Leu Asp Asn Pro Ser
435 440 445
Lys Lys Glu Gln Glu Leu Ile Ala Lys Lys Thr Glu Lys Ala Lys Tyr
450 455 460
Leu Ser Leu Glu Thr Ile Lys Leu Ala Leu Glu Glu Phe Asn Lys His
465 470 475 480
Arg Asp Ile Asp Lys Gln Cys Arg Phe Glu Glu Ile Leu Ala Asn Phe
485 490 495
Ala Ala Ile Pro Met Ile Phe Asp Glu Ile Ala Gln Asn Lys Asp Asn
500 505 510
Leu Ala Gln Ile Ser Ile Lys Tyr Gln Asn Gln Gly Lys Lys Asp Leu
515 520 525
Leu Gln Ala Ser Ala Glu Asp Asp Val Lys Ala Ile Lys Asp Leu Leu
530 535 540
Asp Gln Thr Asn Asn Leu Leu His Lys Leu Lys Ile Phe His Ile Ser
545 550 555 560
Gln Ser Glu Asp Lys Ala Asn Ile Leu Asp Lys Asp Glu His Phe Tyr
565 570 575
Leu Val Phe Glu Glu Cys Tyr Phe Glu Leu Ala Asn Ile Val Pro Leu
580 585 590
Tyr Asn Lys Ile Arg Asn Tyr Ile Thr Gln Lys Pro Tyr Ser Asp Glu
595 600 605
Lys Phe Lys Leu Asn Phe Glu Asn Ser Thr Leu Ala Asn Gly Trp Asp
610 615 620
Lys Asn Lys Glu Pro Asp Asn Thr Ala Ile Leu Phe Ile Lys Asp Asp
625 630 635 640
Lys Tyr Tyr Leu Gly Val Met Asn Lys Lys Asn Asn Lys Ile Phe Asp
645 650 655
Asp Lys Ala Ile Lys Glu Asn Lys Gly Glu Gly Tyr Lys Lys Ile Val
660 665 670
Tyr Lys Leu Leu Pro Gly Ala Asn Lys Met Leu Pro Lys Val Phe Phe
675 680 685
Ser Ala Lys Ser Ile Lys Phe Tyr Asn Pro Ser Glu Asp Ile Leu Arg
690 695 700
Ile Arg Asn His Ser Thr His Thr Lys Asn Gly Ser Pro Gln Lys Gly
705 710 715 720
Tyr Glu Lys Phe Glu Phe Asn Ile Glu Asp Cys Arg Lys Phe Ile Asp
725 730 735
Phe Tyr Lys Gln Ser Ile Ser Lys His Pro Glu Trp Lys Asp Phe Gly
740 745 750
Phe Arg Phe Ser Asp Thr Gln Arg Tyr Asn Ser Ile Asp Glu Phe Tyr
755 760 765
Arg Glu Val Glu Asn Gln Gly Tyr Lys Leu Thr Phe Glu Asn Ile Ser
770 775 780
Glu Ser Tyr Ile Asp Ser Val Val Asn Gln Gly Lys Leu Tyr Leu Phe
785 790 795 800
Gln Ile Tyr Asn Lys Asp Phe Ser Ala Tyr Ser Lys Gly Arg Pro Asn
805 810 815
Leu His Thr Leu Tyr Trp Lys Ala Leu Phe Asp Glu Arg Asn Leu Gln
820 825 830
Asp Val Val Tyr Lys Leu Asn Gly Glu Ala Glu Leu Phe Tyr Arg Lys
835 840 845
Gln Ser Ile Pro Lys Lys Ile Thr His Pro Ala Lys Glu Ala Ile Ala
850 855 860
Asn Lys Asn Lys Asp Asn Pro Lys Lys Glu Ser Val Phe Glu Tyr Asp
865 870 875 880
Leu Ile Lys Asp Lys Arg Phe Thr Glu Asp Lys Phe Phe Phe His Cys
885 890 895
Pro Ile Thr Ile Asn Phe Lys Ser Ser Gly Ala Asn Lys Phe Asn Asp
900 905 910
Glu Ile Asn Leu Leu Leu Lys Glu Lys Ala Asn Asp Val His Ile Leu
915 920 925
Ser Ile Ala Arg Gly Glu Arg His Leu Ala Tyr Tyr Thr Leu Val Asp
930 935 940
Gly Lys Gly Asn Ile Ile Lys Gln Asp Thr Phe Asn Ile Ile Gly Asn
945 950 955 960
Asp Arg Met Lys Thr Asn Tyr His Asp Lys Leu Ala Ala Ile Glu Lys
965 970 975
Asp Arg Asp Ser Ala Arg Lys Asp Trp Lys Lys Ile Asn Asn Ile Lys
980 985 990
Glu Met Lys Glu Gly Tyr Leu Ser Gln Val Val His Glu Ile Ala Lys
995 1000 1005
Leu Val Ile Glu Tyr Asn Ala Ile Val Val Phe Glu Asp Leu Asn
1010 1015 1020
Phe Gly Phe Lys Arg Gly Arg Phe Lys Val Glu Lys Gln Val Tyr
1025 1030 1035
Gln Lys Leu Glu Lys Met Leu Ile Glu Lys Leu Asn Tyr Leu Val
1040 1045 1050
Phe Lys Asp Asn Glu Phe Asp Lys Thr Gly Gly Val Leu Arg Ala
1055 1060 1065
Tyr Gln Leu Thr Ala Pro Phe Glu Thr Phe Lys Lys Met Gly Lys
1070 1075 1080
Gln Thr Gly Ile Ile Tyr Tyr Val Pro Ala Gly Phe Thr Ser Lys
1085 1090 1095
Ile Cys Pro Val Thr Gly Phe Val Asn Gln Leu Tyr Pro Lys Tyr
1100 1105 1110
Glu Ser Val Ser Lys Ser Gln Glu Phe Phe Ser Lys Phe Asp Lys
1115 1120 1125
Ile Cys Tyr Asn Leu Asp Lys Gly Tyr Phe Glu Phe Ser Phe Asp
1130 1135 1140
Tyr Lys Asn Phe Gly Asp Lys Ala Ala Lys Gly Lys Trp Thr Ile
1145 1150 1155
Ala Ser Phe Gly Ser Arg Leu Ile Asn Phe Arg Asn Ser Asp Lys
1160 1165 1170
Asn His Asn Trp Asp Thr Arg Glu Val Tyr Pro Thr Lys Glu Leu
1175 1180 1185
Glu Lys Leu Leu Lys Asp Tyr Ser Ile Glu Tyr Gly His Gly Glu
1190 1195 1200
Cys Ile Lys Ala Ala Ile Cys Gly Glu Ser Asp Lys Lys Phe Phe
1205 1210 1215
Ala Lys Leu Thr Ser Val Leu Asn Thr Ile Leu Gln Met Arg Asn
1220 1225 1230
Ser Lys Thr Gly Thr Glu Leu Asp Tyr Leu Ile Ser Pro Val Ala
1235 1240 1245
Asp Val Asn Gly Asn Phe Phe Asp Ser Arg Gln Ala Pro Lys Asn
1250 1255 1260
Met Pro Gln Asp Ala Asp Ala Asn Gly Ala Tyr His Ile Gly Leu
1265 1270 1275
Lys Gly Leu Met Leu Leu Gly Arg Ile Lys Asn Asn Gln Glu Gly
1280 1285 1290
Lys Lys Leu Asn Leu Val Ile Lys Asn Glu Glu Tyr Phe Glu Phe
1295 1300 1305
Val Gln Asn Arg Asn Asn
1310
<210> 37
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 37
actgcggaaa tttgagcgt 19
<210> 38
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 38
acgctcaaat ttccgcagt 19
<210> 39
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 39
aggcaatggc tgcacatgc 19
<210> 40
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 40
gcatgtgcag ccattgcct 19
<210> 41
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 41
gacgcttggt tctgaggag 19
<210> 42
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 42
ctcctcagaa ccaagcgtc 19
<210> 43
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 43
tccggaaacg cattcctct 19
<210> 44
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 44
agaggaatgc gtttccgga 19
<210> 45
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 45
ccgcgtcagc ccggcccgg 19
<210> 46
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 46
ccgggccggg ctgacgcgg 19
<210> 47
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 47
cgactcccgc tgggcctct 19
<210> 48
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 48
agaggcccag cgggagtcg 19
<210> 49
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 49
ccgttgcgcg ctcgctctc 19
<210> 50
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 50
gagagcgagc gcgcaacgg 19
<210> 51
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 51
ccgcgcatcc tgccaggcc 19
<210> 52
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 52
ggcctggcag gatgcgcgg 19
<210> 53
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 53
ccaacttggc gcgtttcgg 19
<210> 54
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 54
ccgaaacgcg ccaagttgg 19
<210> 55
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 55
accacgcgtc cgagtccgg 19
<210> 56
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 56
ccggactcgg acgcgtggt 19
<210> 57
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 57
tgctcattgt ccctggaca 19
<210> 58
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 58
tgtccaggga caatgagca 19
<210> 59
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 59
ggacaccctg ctcattgtc 19
<210> 60
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 60
gacaatgagc agggtgtcc 19
<210> 61
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 61
accggcagcc tgcgcgtcc 19
<210> 62
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 62
ggacgcgcag gctgccggt 19
<210> 63
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 63
cgatgggcac ccactgctc 19
<210> 64
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 64
gagcagtggg tgcccatcg 19
<210> 65
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 65
ccttcacgtg gacgcgcag 19
<210> 66
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 66
ctgcgcgtcc acgtgaagg 19
<210> 67
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 67
cgtgaaggtg gaagccttc 19
<210> 68
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 68
gaaggcttcc accttcacg 19
<210> 69
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 69
ctccttggtc aggcgccgg 19
<210> 70
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 70
ccggcgcctg accaaggag 19
<210> 71
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 71
tggtcaggcg ccggttccg 19
<210> 72
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 72
cggaaccggc gcctgacca 19
<210> 73
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 73
tagaggtcgc cttctcctc 19
<210> 74
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 74
gaggagaagg cgacctcta 19
<210> 75
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 75
cgacgctcgg gtcgcggtg 19
<210> 76
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 76
caccgcgacc cgagcgtcg 19
<210> 77
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 77
atgctgtcgc cgcgcgggg 19
<210> 78
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 78
ccccgcgcgg cgacagcat 19
<210> 79
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 79
ctcaccctca ccggagcca 19
<210> 80
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 80
tggctccggt gagggtgag 19
<210> 81
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 81
ccgcaaactt tactcctta 19
<210> 82
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 82
taaggagtaa agtttgcgg 19
<210> 83
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 83
ctcctaagat tggcttcac 19
<210> 84
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 84
gtgaagccaa tcttaggag 19
<210> 85
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 85
ccggagccac tcctaagat 19
<210> 86
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 86
atcttaggag tggctccgg 19
<210> 87
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 87
ttctctaccc tacgtctca 19
<210> 88
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 88
tgagacgtag ggtagagaa 19
<210> 89
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 89
tacgtctcat tctccgcaa 19
<210> 90
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 90
ttgcggagaa tgagacgta 19
<210> 91
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 91
gctaggcctc cagcccttc 19
<210> 92
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 92
gaagggctgg aggcctagc 19
<210> 93
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 93
acaggtggcg ccgcaactt 19
<210> 94
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 94
aagttgcggc gccacctgt 19
<210> 95
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 95
agccggaggc gcgagagtc 19
<210> 96
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид
<400> 96
gactctcgcg cctccggct 19
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| ЛЕЧЕНИЕ РАКА С ИСПОЛЬЗОВАНИЕМ ХИМЕРНОГО АНТИГЕНСПЕЦИФИЧЕСКОГО РЕЦЕПТОРА НА ОСНОВЕ ГУМАНИЗИРОВАННОГО АНТИТЕЛА ПРОТИВ EGFRvIII | 2014 |
|
RU2831418C2 |
| ГЛИКАНЗАВИСИМЫЕ ИММУНОТЕРАПЕВТИЧЕСКИЕ МОЛЕКУЛЫ | 2016 |
|
RU2754661C2 |
| КОМПОЗИЦИИ И СПОСОБЫ ДЛЯ ИЗБИРАТЕЛЬНОЙ ЭКСПРЕССИИ БЕЛКА | 2017 |
|
RU2795467C2 |
| КОНСТРУКЦИИ ДНК-АНТИТЕЛ ДЛЯ ПРИМЕНЕНИЯ ПРОТИВ БОЛЕЗНИ ЛАЙМА | 2017 |
|
RU2813829C2 |
| ХИМЕРНЫЙ АНТИГЕННЫЙ РЕЦЕПТОР (CAR) ПРОТИВ CD123 ДЛЯ ИСПОЛЬЗОВАНИЯ В ЛЕЧЕНИИ ЗЛОКАЧЕСТВЕННЫХ ОПУХОЛЕЙ | 2015 |
|
RU2724999C2 |
| ЛЕЧЕНИЕ ЗЛОКАЧЕСТВЕННОГО НОВООБРАЗОВАНИЯ С ИСПОЛЬЗОВАНИЕМ ГУМАНИЗИРОВАННОГО ХИМЕРНОГО АНТИГЕННОГО РЕЦЕПТОРА ПРОТИВ ВСМА | 2015 |
|
RU2751660C2 |
| T-КЛЕТОЧНЫЕ РЕЦЕПТОРЫ, РАСПОЗНАЮЩИЕ МУТАЦИЮ R175H ИЛИ Y220C В P53 | 2020 |
|
RU2830061C2 |
| МОДИФИЦИРОВАННЫЕ НЕПРИРОДНЫЕ ЛИГАНДЫ NKG2D, КОТОРЫЕ ИЗБИРАТЕЛЬНО ДОСТАВЛЯЮТ ПРИСОЕДИНЕННЫЕ ГЕТЕРОЛОГИЧНЫЕ МОЛЕКУЛЫ К НЕПРИРОДНЫМ РЕЦЕПТОРАМ NKG2D НА CAR-КЛЕТКАХ | 2020 |
|
RU2823728C2 |
| ВАКЦИНА И СПОСОБЫ ОБНАРУЖЕНИЯ И ПРОФИЛАКТИКИ ФИЛЯРИОЗА | 2021 |
|
RU2832185C1 |
| ТЕРАПЕВТИЧЕСКИЕ СРЕДСТВА И СПОСОБЫ ДЛЯ ЛЕЧЕНИЯ TLR2-ОПОСРЕДОВАННЫХ ЗАБОЛЕВАНИЙ И НАРУШЕНИЙ | 2019 |
|
RU2791022C2 |

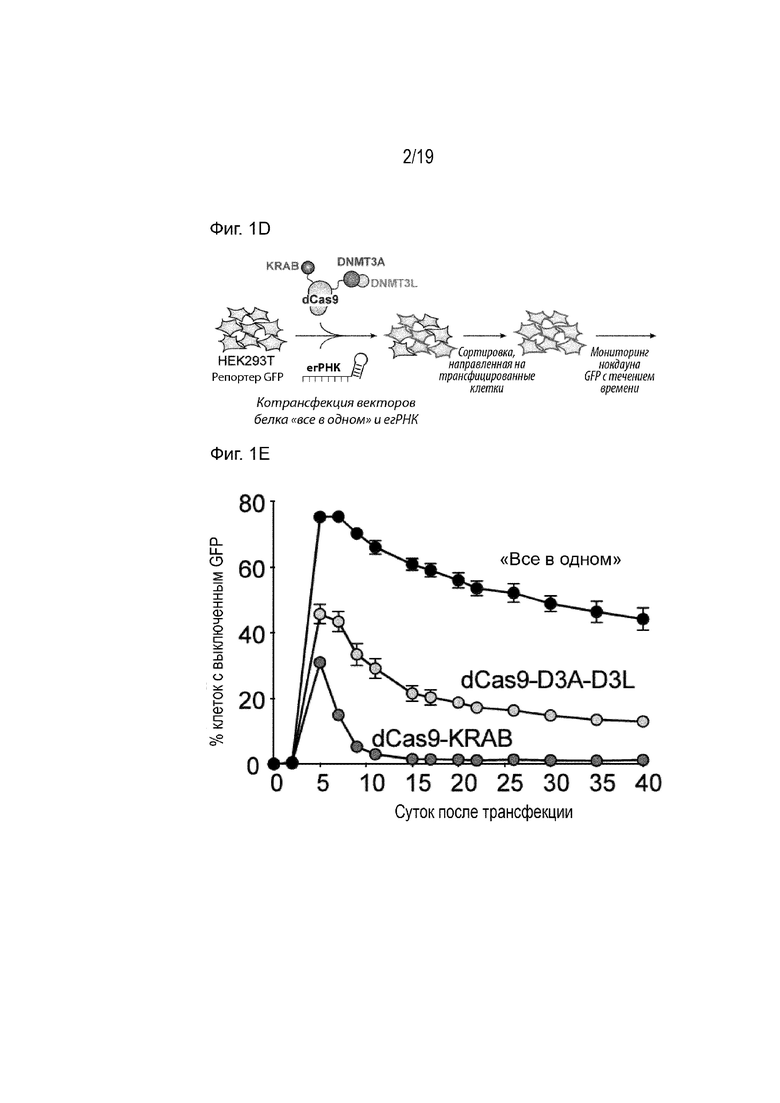

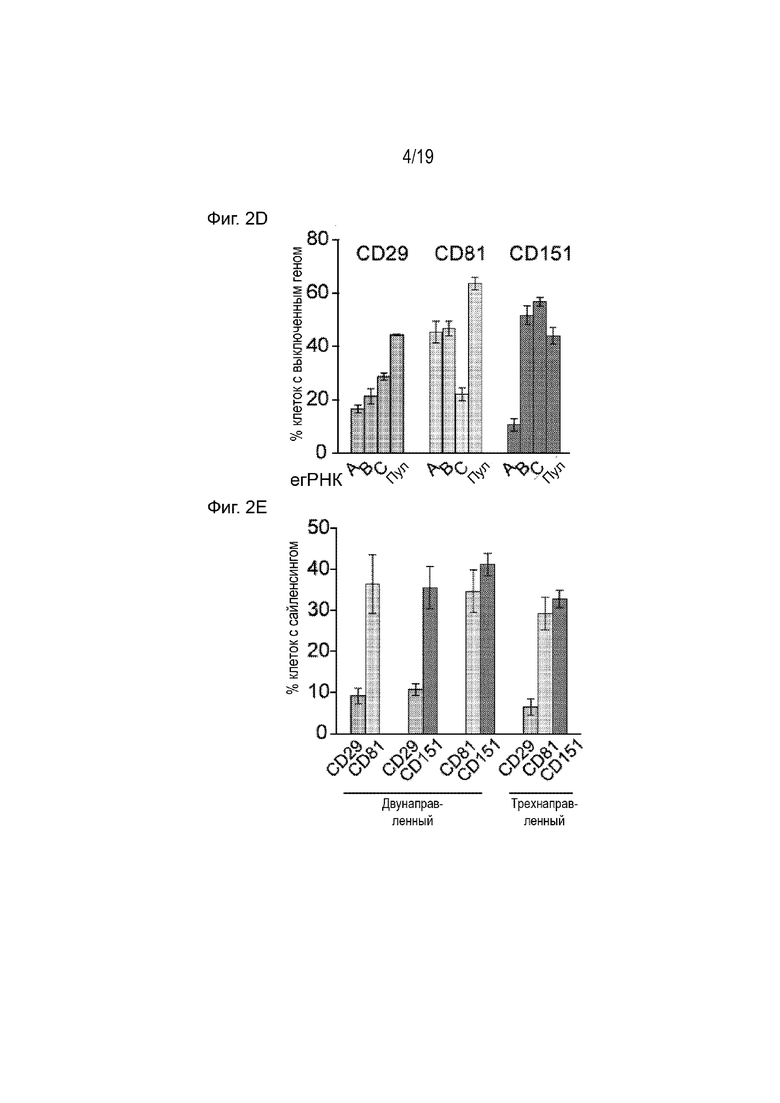
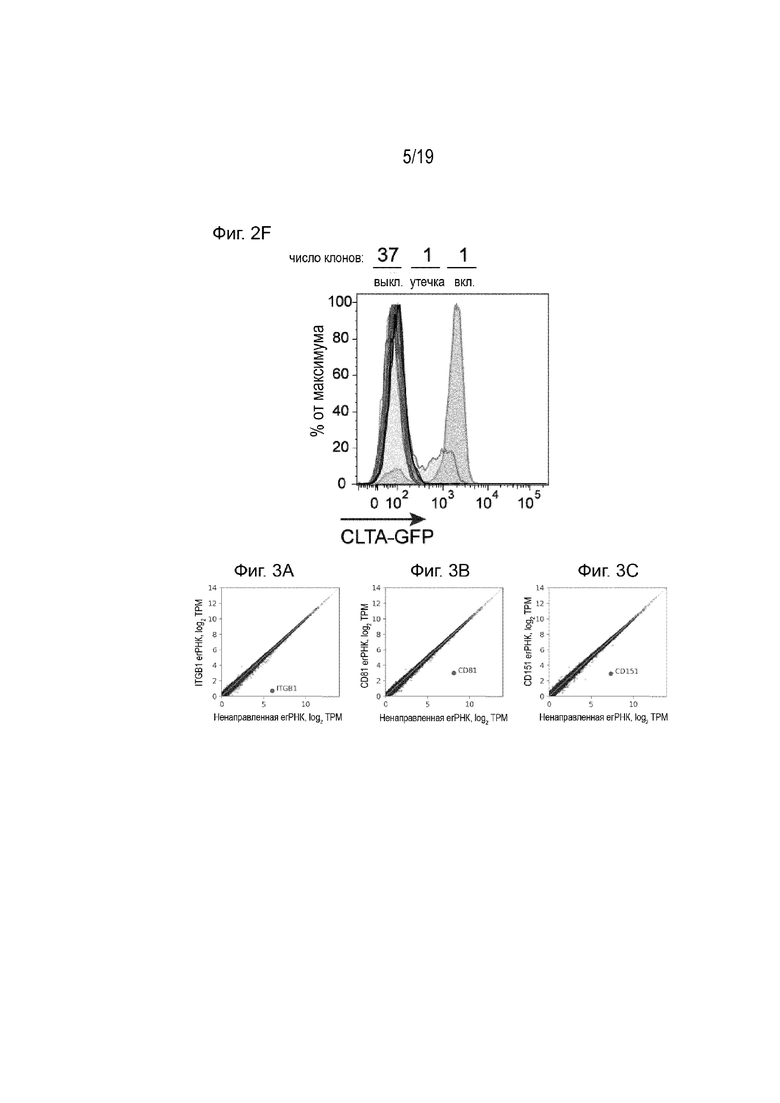


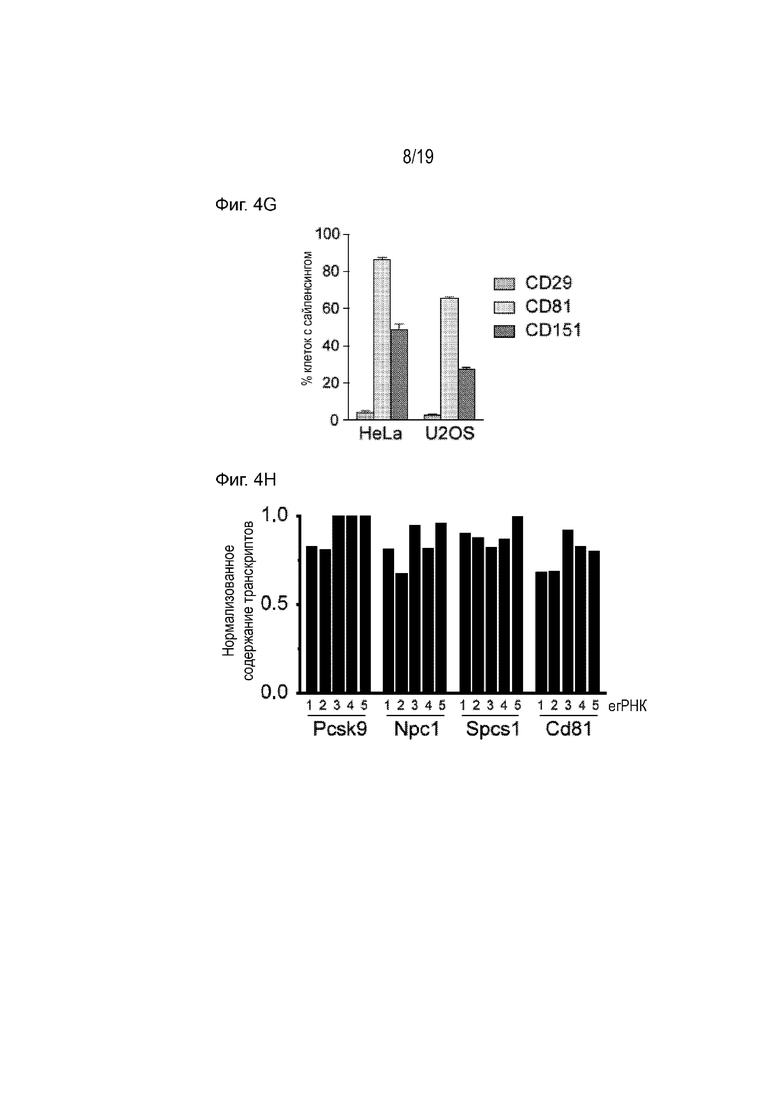


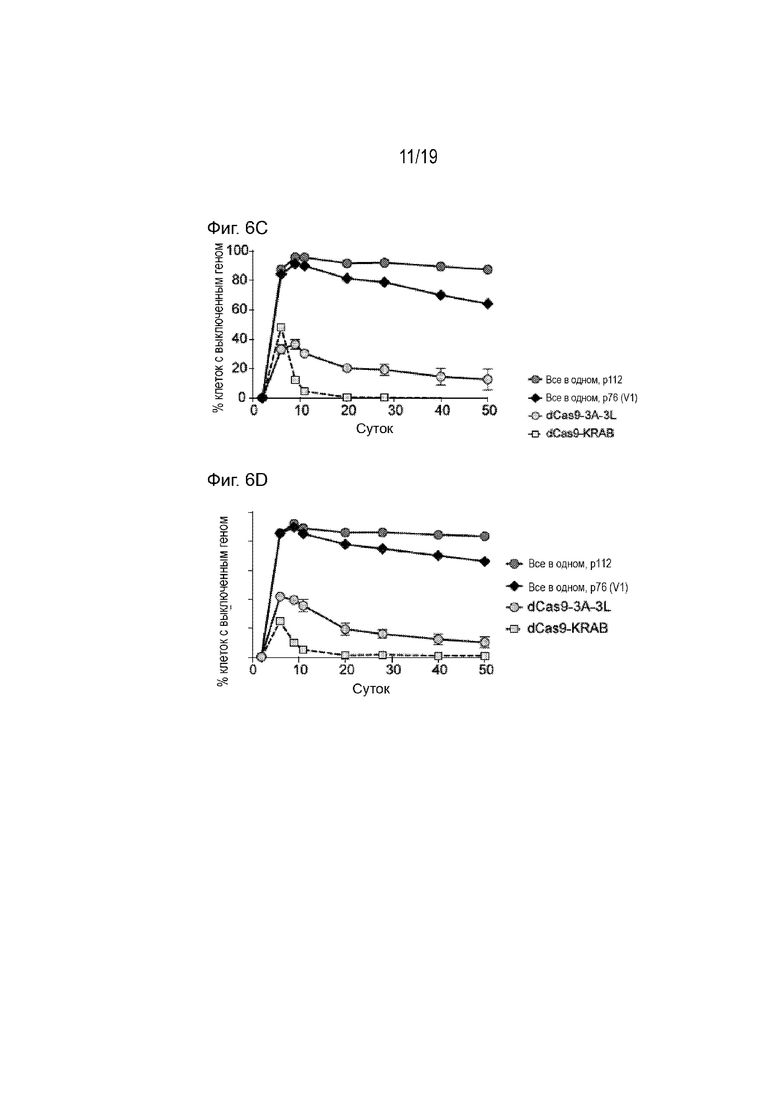

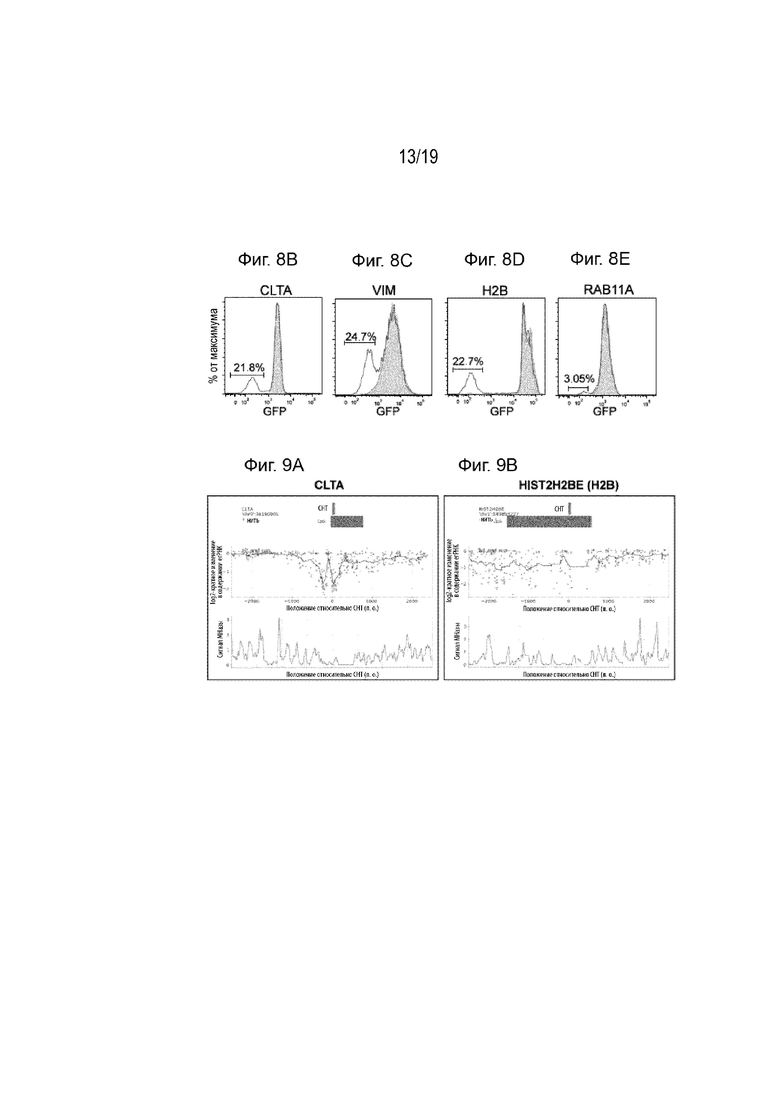

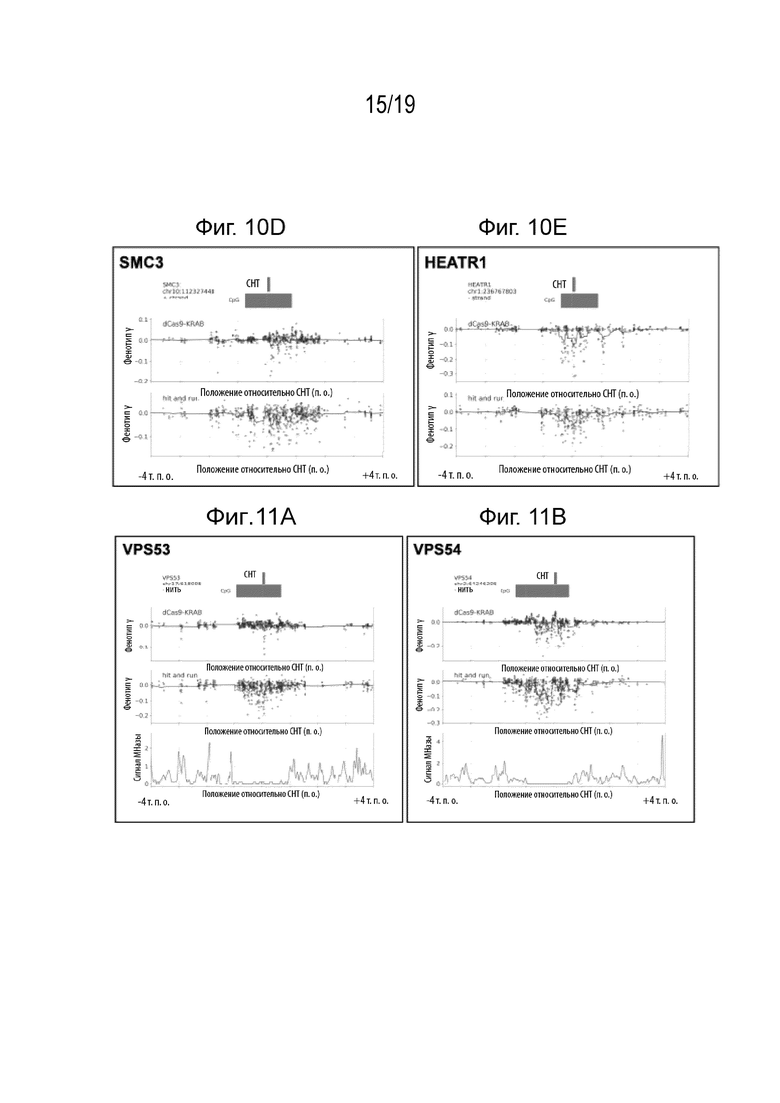
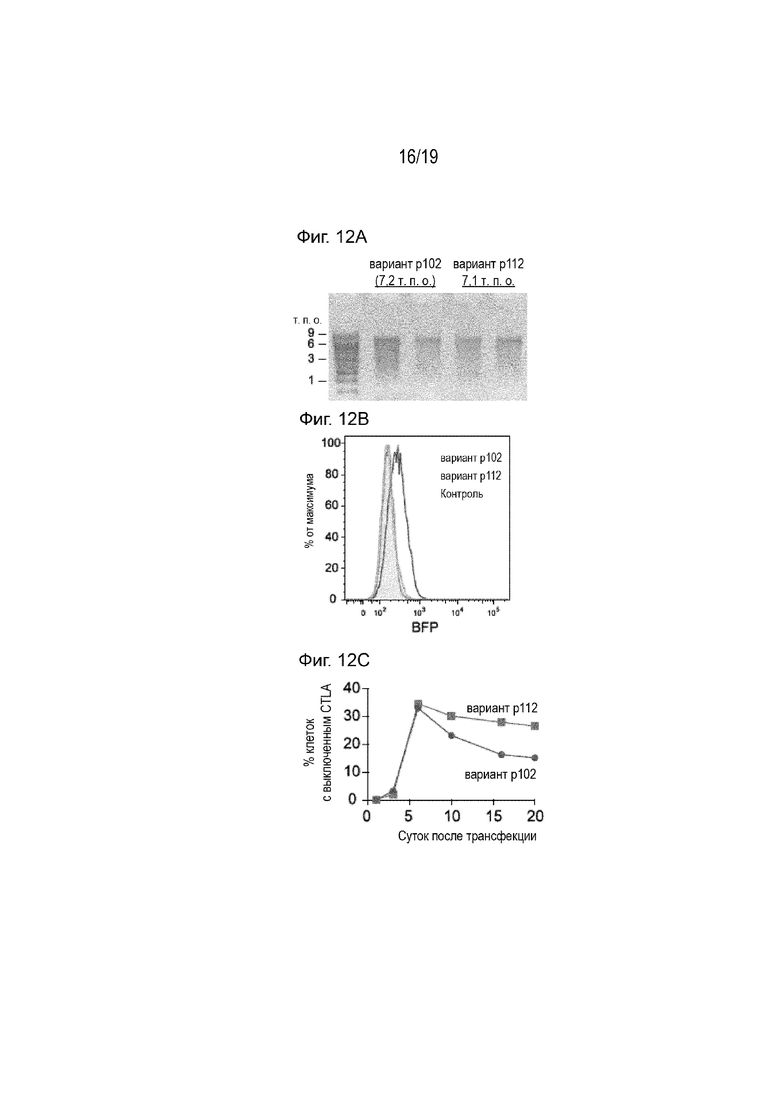
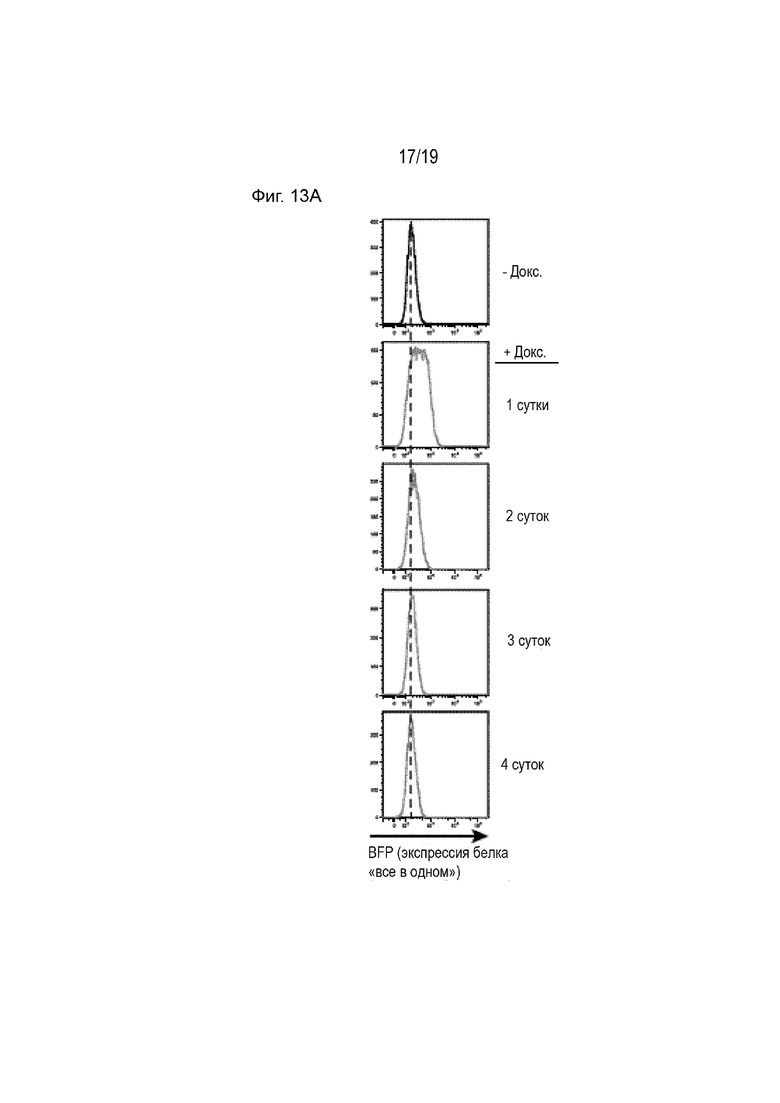
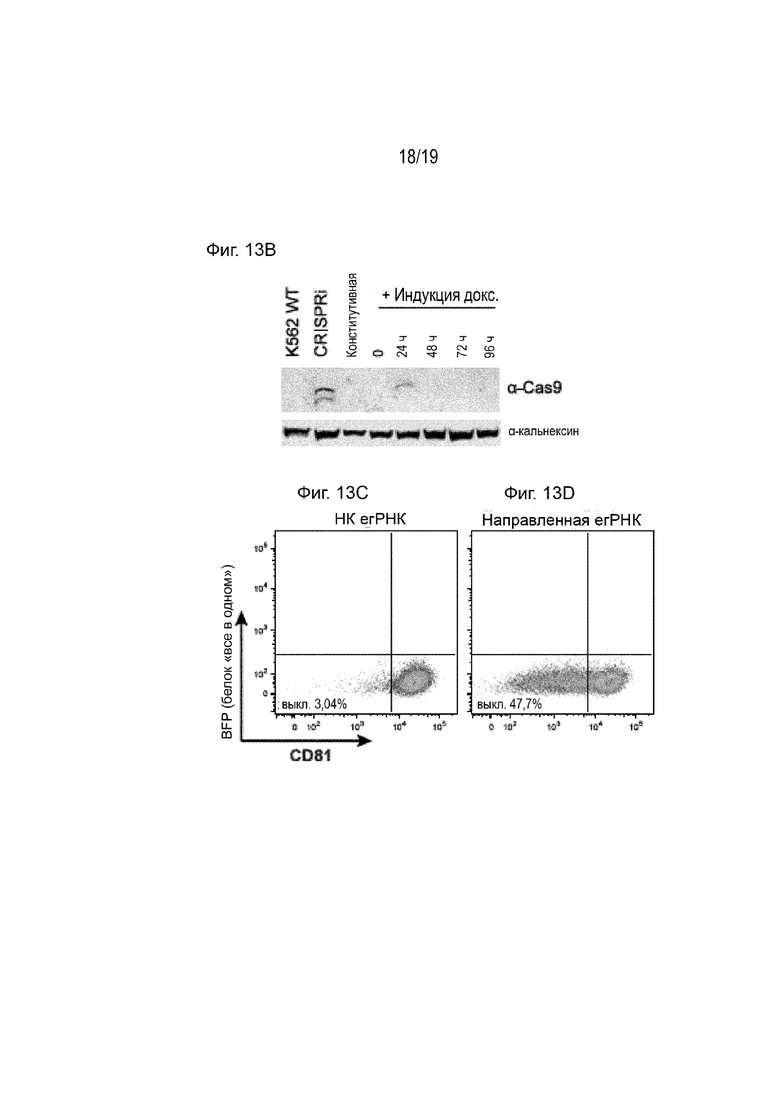
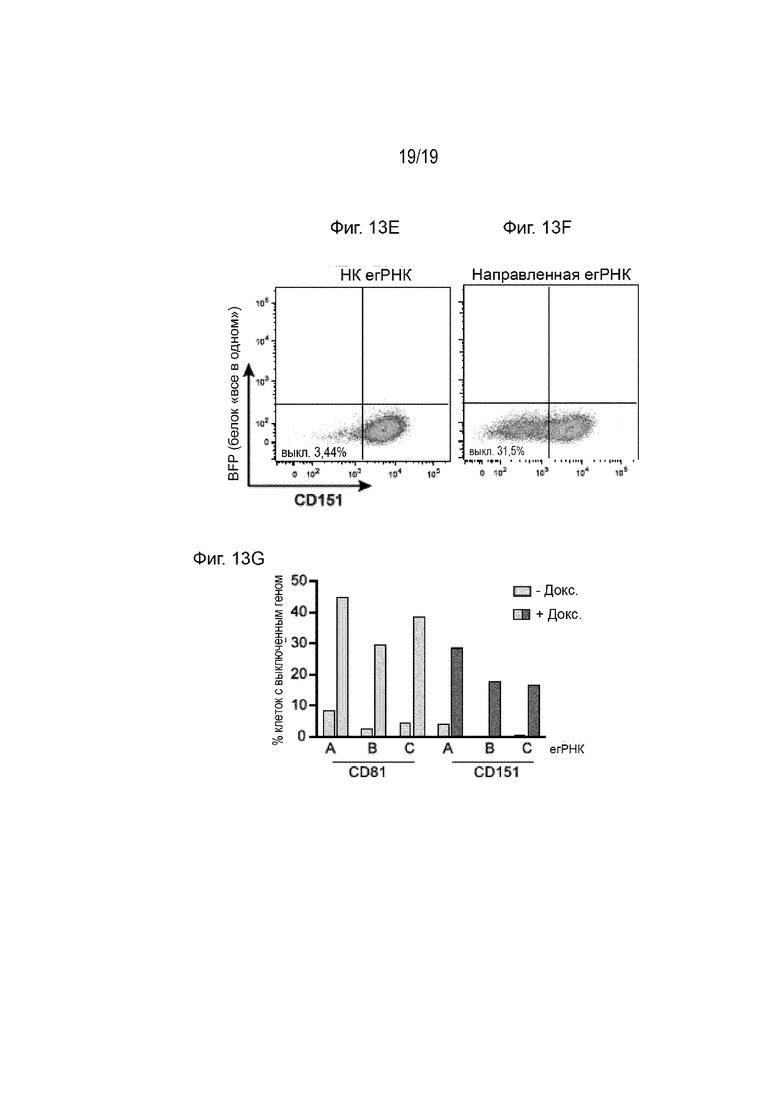
Изобретение относится к области биотехнологии, в частности к слитому белку, содержащему в направлении от N-конца до С-конца домен метилтрансферазы ДНК, фермент нуклеазо-дефицитную РНК-направляемую эндонуклеазу ДНК и домен, связанный с боксом  или домен, связанный с боксом
или домен, связанный с боксом  , фермент нуклеазо-дефицитную РНК-направляемую эндонуклеазу ДНК и домен метилтрансферазы ДНК, а также комплекс, его содержащий. Также раскрыты молекула нуклеиновой кислоты, кодирующая вышеуказанный слитый белок, а также вектор и клетка, ее содержащие. Изобретение эффективно для сайленсинга последовательности целевой нуклеиновой кислоты в клетке. 8 н. и 52 з.п. ф-лы, 13 ил., 2 табл., 12 пр.
, фермент нуклеазо-дефицитную РНК-направляемую эндонуклеазу ДНК и домен метилтрансферазы ДНК, а также комплекс, его содержащий. Также раскрыты молекула нуклеиновой кислоты, кодирующая вышеуказанный слитый белок, а также вектор и клетка, ее содержащие. Изобретение эффективно для сайленсинга последовательности целевой нуклеиновой кислоты в клетке. 8 н. и 52 з.п. ф-лы, 13 ил., 2 табл., 12 пр.
1. Слитый белок для сайленсинга последовательности целевой нуклеиновой кислоты, содержащий, в направлении от N-конца до С-конца:
(i) домен метилтрансферазы ДНК, фермент нуклеазо-дефицитную РНК-направляемую эндонуклеазу ДНК и домен, связанный с боксом  ; или
; или
(ii) домен, связанный с боксом  , фермент нуклеазо-дефицитную РНК-направляемую эндонуклеазу ДНК и домен метилтрансферазы ДНК.
, фермент нуклеазо-дефицитную РНК-направляемую эндонуклеазу ДНК и домен метилтрансферазы ДНК.
2. Слитый белок по п. 1, отличающийся тем, что указанный фермент нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК представляет собой нуклеазо-дефицитную эндонуклеазу CRISPR класса II.
3. Слитый белок по п. 1, отличающийся тем, что указанный домен метилтрансферазы ДНК содержит белок Dnmt3A и белок Dnmt3L (домен Dnmt3A-3L).
4. Слитый белок по п. 1, отличающийся тем, что указанный слитый белок включает в себя, в направлении от N-конца до С-конца, домен метилтрансферазы ДНК, фермент нуклеазо-дефицитную РНК-направляемую эндонуклеазу ДНК и домен, связанный с боксом  .
.
5. Слитый белок по п. 4, отличающийся тем, что указанный фермент нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК представляет собой dCas9, а указанный домен метилтрансферазы ДНК содержит белок Dnmt3A и белок Dnmt3L (домен Dnmt3A-3L); где указанный dCas9 ковалентно связан с доменом Dnmt3A-3L через линкер XTEN.
6. Слитый белок по п. 5, отличающийся тем, что указанный dCas9 ковалентно связан с указанным доменом Dnmt3A-3L через линкер XTEN, и при этом указанный домен Dnmt3A-3L ковалентно связан с указанным доменом, связанным с боксом  , через линкер XTEN.
, через линкер XTEN.
7. Слитый белок по п. 1, отличающийся тем, что указанный слитый белок включает в себя, в направлении от N-конца до С-конца, домен, связанный с боксом  , фермент нуклеазо-дефицитную РНК-направляемую эндонуклеазу ДНК, линкер XTEN и домен метилтрансферазы ДНК.
, фермент нуклеазо-дефицитную РНК-направляемую эндонуклеазу ДНК, линкер XTEN и домен метилтрансферазы ДНК.
8. Слитый белок по п. 7, отличающийся тем, что указанный фермент нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК представляет собой dCas9, а указанный домен метилтрансферазы ДНК содержит белок Dnmt3A и белок Dnmt3L (домен Dnmt3A-3L).
9. Слитый белок по п. 8, отличающийся тем, что указанный dCas9 ковалентно связан с указанным доменом Dnmt3A-3L через линкер XTEN, и при этом указанный домен, связанный с боксом  , ковалентно связан с указанным dCas9 через линкер XTEN.
, ковалентно связан с указанным dCas9 через линкер XTEN.
10. Слитый белок по п. 1, отличающийся тем, что указанный фермент нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК ковалентно связан с указанным доменом, связанным с боксом  , через пептидный линкер.
, через пептидный линкер.
11. Слитый белок по п. 1, отличающийся тем, что указанный фермент нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК ковалентно связан с указанным доменом метилтрансферазы ДНК через пептидный линкер.
12. Слитый белок по п. 1, отличающийся тем, что указанный домен, связанный с боксом  , ковалентно связан с указанным доменом метилтрансферазы ДНК через линкер XTEN.
, ковалентно связан с указанным доменом метилтрансферазы ДНК через линкер XTEN.
13. Слитый белок по п. 10, отличающийся тем, что указанный пептидный линкер представляет собой линкер XTEN.
14. Слитый белок по п. 13, отличающийся тем, что указанный линкер XTEN содержит от около 16 до около 80 аминокислотных остатков.
15. Слитый белок по п. 1, дополнительно содержащий сигнальный пептид ядерной локализации.
16. Слитый белок по п. 1, отличающийся тем, что указанный слитый белок содержит аминокислотную последовательность с SEQ ID NO: 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14 или 15.
17. Нуклеиновая кислота, кодирующая слитый белок по п. 1.
18. Нуклеиновая кислота по п. 17, отличающаяся тем, что указанная последовательность нуклеиновой кислоты представляет собой информационную РНК.
19. Комплекс для сайленсинга последовательности целевой нуклеиновой кислоты, содержащий: (i) слитый белок по п. 1; и (ii) полинуклеотид, содержащий: (а) нацеливающую на ДНК последовательность, комплементарную целевой полинуклеотидной последовательности; и (b) последовательность связывания для указанного фермента нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК, при этом указанный фермент нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК связан с указанным полинуклеотидом через связывающую последовательность.
20. Комплекс по п. 19, отличающийся тем, что указанная целевая полинуклеотидная последовательность представляет собой часть гена.
21. Комплекс по п. 19, отличающийся тем, что указанная целевая полинуклеотидная последовательность представляет собой часть последовательности, регулирующей транскрипцию.
22. Комплекс по п. 19, отличающийся тем, что указанная целевая полинуклеотидная последовательность представляет собой часть промотора, энхансера или сайленсера.
23. Комплекс по п. 19, отличающийся тем, что указанная целевая полинуклеотидная последовательность находится в пределах около 3000 п.о. и фланкирует сайт начала транскрипции.
24. Экспрессионный вектор для сайленсинга целевого гена, содержащий последовательность нуклеиновой кислоты, кодирующую слитый белок по п. 1.
25. Экспрессионный вектор по п. 24, дополнительно содержащий полинуклеотид, отличающийся тем, что указанный полинуклеотид содержит: (а) нацеливающую на ДНК последовательность, которая комплементарна целевой полинуклеотидной последовательности; и (b) последовательность связывания для фермента нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК.
26. Выделенная клетка для направленного сайленсинга гена, содержащая слитый белок по п. 1; нуклеиновую кислоту по п. 17; комплекс по п. 19 или вектор по п. 24, где указанная клетка содержит целевой ген, подвергшийся сайленсингу.
27. Выделенная клетка по п. 26, отличающаяся тем, что указанная клетка представляет собой эукариотическую клетку.
28. Выделенная клетка по п. 26, отличающаяся тем, что указанная клетка представляет собой клетку млекопитающего.
29. Способ сайленсинга последовательности целевой нуклеиновой кислоты в клетке, включающий: (i) доставку первого полинуклеотида, кодирующего слитый белок по п. 1 в клетку, содержащую указанную целевую нуклеиновую кислоту; и (ii) доставку в указанную клетку второго полинуклеотида, который включает в себя (a) нацеленную на ДНК последовательность, которая комплементарна указанной последовательности целевой нуклеиновой кислоты, и (b) связывающую последовательность для указанного фермента нуклеазо-дефицитная РНК-направляемая эндонуклеаза ДНК.
30. Способ по п. 29, отличающийся тем, что указанный слитый белок вызывает сайленсинг указанной последовательности целевой нуклеиновой кислоты в указанной клетке путем метилирования хроматина, содержащего указанную последовательность целевой нуклеиновой кислоты, и (или) путем введения репрессивных хроматиновых маркеров в хроматин, содержащий указанную последовательность целевой нуклеиновой кислоты.
31. Способ по п. 29, отличающийся тем, что указанный первый полинуклеотид содержится в первом векторе.
32. Способ по п. 29, отличающийся тем, что указанный первый полинуклеотид содержится во втором векторе.
33. Способ по п. 32, отличающийся тем, что указанные первый вектор и второй вектор одинаковы.
34. Способ по п. 32, отличающийся тем, что указанный первый вектор отличается от указанного второго вектора.
35. Способ по п. 29, отличающийся тем, что указанная клетка представляет собой клетку млекопитающего.
36. Способ по п. 29, отличающийся тем, что специфичность указанного способа в 2 раза выше, чем специфичность по отношению к последовательности нецелевой нуклеиновой кислоты.
37. Способ сайленсинга последовательности целевой нуклеиновой кислоты в клетке, включающий в себя доставку комплекса по п. 19 в клетку, содержащую указанную целевую нуклеиновую кислоту.
38. Способ по п. 37, отличающийся тем, что указанный комплекс вызывает сайленсинг указанной последовательности целевой нуклеиновой кислоты в указанной клетке путем метилирования хроматина, содержащего указанную последовательность целевой нуклеиновой кислоты, и (или) путем введения репрессивных хроматиновых маркеров в хроматин, содержащий указанную последовательность целевой нуклеиновой кислоты.
39. Способ по п. 37, отличающийся тем, что указанная клетка представляет собой клетку млекопитающего.
40. Способ по п. 37, отличающийся тем, что специфичность указанного способа в 2 раза выше, чем специфичность по отношению к последовательности нецелевой нуклеиновой кислоты.
41. Слитый белок по п. 1, отличающийся тем, что указанный фермент нуклеазо-дефицитной РНК-направляемой эндонуклеазы ДНК представляет собой фермент dCas9, который содержит аминокислотную последовательность, имеющую по меньшей мере 85% идентичности последовательности с SEQ ID NO:23; ddCas12a, который содержит аминокислотную последовательность, по меньшей мере на 85% идентичную SEQ ID NO:34; dLbCfp1, который содержит аминокислотную последовательность, по меньшей мере на 85% идентичную SEQ ID NO:35; или dFnCfp1, который содержит аминокислотную последовательность, по меньшей мере на 85% идентичную SEQ ID NO: 36.
42. Слитый белок по п. 1, отличающийся тем, что указанный домен, связанный с боксом  , содержит аминокислотную последовательность, имеющую по меньшей мере 85% идентичности последовательности с SEQ ID NO: 16.
, содержит аминокислотную последовательность, имеющую по меньшей мере 85% идентичности последовательности с SEQ ID NO: 16.
43. Слитый белок по п. 3, отличающийся тем, что указанный белок Dnmt3A содержит аминокислотную последовательность, имеющую по меньшей мере 85% идентичности последовательности с SEQ ID NO: 26, а указанный белок Dnmt3L содержит аминокислотную последовательность, имеющую по меньшей мере 85% идентичности последовательности с SEQ ID NO: 28.
44. Слитый белок по п. 1, отличающийся тем, что указанный слитый белок содержит аминокислотную последовательность, имеющую по меньшей мере 85% идентичности последовательности с SEQ ID NO: 15.
45. Слитый белок по п. 1, отличающийся тем, что указанный фермент нуклеазо-дефицитной РНК-направляемой эндонуклеазы ДНК выбран из группы, состоящей из dCas9, ddCpf1, ddCas12a, ddLbCfp1 и ddFnCfp1.
46. Слитый белок для сайленсинга последовательности целевой нуклеиновой кислоты, содержащий домен метилтрансферазы ДНК, фермент нуклеазо-дефицитную РНК-направляемую эндонуклеазу ДНК и домен, связанный с боксом  ; где
; где
(1) указанный фермент нуклеазо-дефицитной РНК-направляемой эндонуклеазы ДНК представляет собой фермент dCas9, который содержит аминокислотную последовательность, имеющую по меньшей мере 85% идентичности последовательности с SEQ ID NO:23; ddCas12a, который содержит аминокислотную последовательность, по меньшей мере на 85% идентичную SEQ ID NO:34; dLbCfp1, который содержит аминокислотную последовательность, по меньшей мере на 85% идентичную SEQ ID NO:35; или dFnCfp1, который содержит аминокислотную последовательность, по меньшей мере на 85% идентичную SEQ ID NO: 36;
(2) указанный домен, связанный с боксом  , содержит аминокислотную последовательность, имеющую по меньшей мере 85% идентичности последовательности с SEQ ID NO: 16; и
, содержит аминокислотную последовательность, имеющую по меньшей мере 85% идентичности последовательности с SEQ ID NO: 16; и
(3) домен ДНК-метилтрансферазы содержит белок Dnmt3A, белок Dnmt3L или белок Dnmt3A и белок Dnmt3L; при этом указанный белок Dnmt3A содержит аминокислотную последовательность, имеющую по меньшей мере 85% идентичности последовательности с SEQ ID NO: 26, а указанный белок Dnmt3L содержит аминокислотную последовательность, имеющую по меньшей мере 85% идентичности последовательности с SEQ ID NO: 28.
47. Слитый белок по п. 46, отличающийся тем, что указанный домен, связанный с боксом  , содержит аминокислотную последовательность, имеющую по меньшей мере 90% идентичности последовательности с SEQ ID NO: 16.
, содержит аминокислотную последовательность, имеющую по меньшей мере 90% идентичности последовательности с SEQ ID NO: 16.
48. Слитый белок по п. 47, отличающийся тем, что указанный домен, связанный с боксом  , содержит аминокислотную последовательность, имеющую по меньшей мере 95% идентичности последовательности с SEQ ID NO: 16.
, содержит аминокислотную последовательность, имеющую по меньшей мере 95% идентичности последовательности с SEQ ID NO: 16.
49. Слитый белок по п. 48, отличающийся тем, что указанный домен, связанный с боксом  , содержит аминокислотную последовательность, имеющую по меньшей мере 99% идентичности последовательности с SEQ ID NO: 16.
, содержит аминокислотную последовательность, имеющую по меньшей мере 99% идентичности последовательности с SEQ ID NO: 16.
50. Слитый белок по п. 46, отличающийся тем, что указанный белок Dnmt3A содержит аминокислотную последовательность, имеющую по меньшей мере 90% идентичности последовательности с SEQ ID NO: 26.
51. Слитый белок по п. 50, отличающийся тем, что указанный белок Dnmt3A содержит аминокислотную последовательность, имеющую по меньшей мере 95% идентичности последовательности с SEQ ID NO: 26.
52. Слитый белок по п. 51, отличающийся тем, что указанный белок Dnmt3A содержит аминокислотную последовательность, имеющую по меньшей мере 99% идентичности последовательности с SEQ ID NO: 26.
53. Слитый белок по п. 46, отличающийся тем, что указанный белок Dnmt3L содержит аминокислотную последовательность, имеющую по меньшей мере 90% идентичности последовательности с SEQ ID NO: 28.
54. Слитый белок по п. 53, отличающийся тем, что указанный белок Dnmt3L содержит аминокислотную последовательность, имеющую по меньшей мере 95% идентичности последовательности с SEQ ID NO: 28.
55. Слитый белок по п. 54, отличающийся тем, что указанный белок Dnmt3L содержит аминокислотную последовательность, имеющую по меньшей мере 99% идентичности последовательности с SEQ ID NO: 28.
56. Слитый белок по п. 46, отличающийся тем, что указанный dCas9 содержит аминокислотную последовательность, имеющую по меньшей мере 90% идентичности последовательности с SEQ ID NO: 23.
57. Слитый белок по п. 56, отличающийся тем, что указанный dCas9 содержит аминокислотную последовательность, имеющую по меньшей мере 95% идентичности последовательности с SEQ ID NO: 23.
58. Слитый белок по п. 57, отличающийся тем, что указанный dCas9 содержит аминокислотную последовательность, имеющую по меньшей мере 99% идентичности последовательности с SEQ ID NO: 23.
59. Слитый белок по п. 46, отличающийся тем, что указанный домен, связанный с боксом  , содержит аминокислотную последовательность SEQ ID NO: 16, указанный белок Dnmt3A содержит аминокислотную последовательность SEQ ID NO: 26, указанный белок Dnmt3L содержит аминокислотную последовательность SEQ ID NO: 28, а указанный dCas9 содержит аминокислотную последовательность SEQ ID NO: 23.
, содержит аминокислотную последовательность SEQ ID NO: 16, указанный белок Dnmt3A содержит аминокислотную последовательность SEQ ID NO: 26, указанный белок Dnmt3L содержит аминокислотную последовательность SEQ ID NO: 28, а указанный dCas9 содержит аминокислотную последовательность SEQ ID NO: 23.
60. Слитый белок по п. 59, отличающийся тем, что указанный слитый белок содержит, в направлении от N-конца до С-конца, домен метилтрансферазы ДНК, фермент нуклеазо-дефицитную РНК-направляемую эндонуклеазу ДНК и домен, связанный с боксом  .
.
| WO2016063264 A1, 28.04.2016 | |||
| TAFADZWA MLAMBO et al., Designer epigenome modifiers enable robust and sustained gene silencing in clinically relevant human cells, Nucleic Acids Research, 10 March 2018, Vol | |||
| Способ изготовления звездочек для французской бороны-катка | 1922 |
|
SU46A1 |
| Разборный с внутренней печью кипятильник | 1922 |
|
SU9A1 |
| СЧЕТНЫЙ ПРИБОР | 1926 |
|
SU4456A1 |
| WO2018053035 A1, 22.03.2018 | |||
| JOHN P GUILINGER et al., Fusion of catalytically inactive Cas9 to FokI nuclease improves | |||
Авторы
Даты
2023-10-03—Публикация
2019-04-19—Подача