Изобретение относится к гену Н5, кодирующему стабилизированную форму белка гемагглютинина вируса гриппа A (H5N8), рекомбинантной плазмидной ДНК pVAX-H5, содержащая указанный ген Н5 и стабилизированной форме белка гемагглютинина, продуцируемой рекомбинантной плазмидной ДНК pVAX-H5 в клетках эукариот (организме животных) и индуцирующему специфический иммунный ответ против высокопатогенного вируса гриппа A (H5N8), и может быть использовано в области биотехнологии, медицины и иммунологии. Плазмида pVAX-H5 индуцирует НА/Н5-специфический гуморальный и клеточный ответ у лабораторных животных и обеспечивает защиту мышей линии Balb/c от заболевания, вызванного штаммом вируса гриппа A/Astrakhan/3212/2020 (H5N8). Последние два десятилетия документируется все больше случаев инфицирования людей вирусами гриппа птиц с высоким уровнем летальности. Есть вероятность, что данные вирусы приобретут способность передаваться от человека к человеку, что может привести к новой пандемии [1]. В России впервые выявлены случаи заражения людей птичьим гриппом A (H5N8) в 2020 году. В мае 2023 года в Москве был объявлен карантин из-за вспышек птичьего гриппа A (H5N1). Появляющиеся штаммы вируса гриппа усиливают острую потребность в вакцинных технологиях, позволяющих быстро производить препарат вакцины в больших объемах и индуцировать специфический иммунный ответ. Подходящей платформой для индукции клеточного и гуморального иммунитета являются вакцины на основе нуклеиновых кислот, в частности ДНК [2]. ДНК-вакцины обладают следующими преимуществами: неинфекционны и легко нарабатываются в больших количествах, сохраняют свою стабильность в широком диапазоне температур; при введении ДНК-вакцины не индуцируется иммунитет против плазмиды-носителя; способны обеспечить индукцию Т-клеточного иммунитета за счет синтеза белка внутри клетки, что приводит к естественной презентации фрагментов эндогенных белков молекулами МНС I и II класса.
Наиболее близким аналогом (прототипом) является ДНК-вакцина, кодирующая гемагглютинин Н5 в составе векторной плазмиды pCAGGK (патент CN106039303, МПК А61К39/145, опубл. 26.10.2016 г.). Ген Н5 в данной вакцине соответствует последовательности гемагглютинина штамма A/chicken/Guangdong/E85/2011. Данный ген оптимизирован для увеличения экспрессии в клетках кур и клонирован в составе экспрессионного вектора, содержащего ген устойчивости к канамицину. Экспрессия целевого гена была показана на клетках НЕК293Т in vitro. Двукратная внутримышечная иммунизация цыплят привела к формированию гуморального иммунитета: сыворотки иммунизированных цыплят были способны ингибировать гемагглютинацию эритроцитов в РТГА. Однако данная вакцинная конструкция создана для предотвращения вспышек заболевания, вызванного высокопатогенным вирусом птичьего гриппа с высоким уровнем летальности (H5Nx), среди птиц, и не предполагает ее использования для млекопитающих.
Техническим результатом заявляемого изобретения является создание безопасной и эффективной ДНК-вакцины, включающей ген гемагглютинина вируса гриппа A (H5N8), способной индуцировать специфический гуморальный и Т-клеточный иммунитет против вируса гриппа A (H5N8).
Указанный технический результат достигается тем, что создан ген, кодирующий стабилизированную форму белка гемагглютинина вируса гриппа А (H5N8), имеющий последовательность SEQ ID NO: 2 размером 1682 п.н. и содержащий на 5'-конце последовательность Козак (GCCGCCACCATGGAG) и сайт для эндонуклеазы рестрикции AsuNHI, а на 3'-конце - два стоп-кодона (TAATGA) и сайт для эндонуклеазы рестрикции CciNI.
Указанный технический результат достигается также тем, что сконструирована рекомбинантная плазмидная ДНК pVAX-H5, имеющая нуклеотидную последовательность SEQ ID NO: 1 размером 4590 п.н., молекулярный вес 2838763 Да, содержащая целевой ген Н5 по п. 1, кодирующий стабилизированную форму белка гемагглютинина вируса гриппа A (H5N8), перед которым находится энхансер и промотор CMV, обеспечивающий транскрипцию гена в эукариотической системе, и состоящая из следующих фрагментов: векторный фрагмент 2367-710 плазмиды pVAX размером 2934 п.н., содержащий bGH поли(А) сигнал и имеющий координаты с 2420 по 2644 п.н.; ген устойчивости к неомицину/канамицину NeoR/KanR размером 795 п.н., имеющий координаты с 2817 по 3611 п.н.; точку начала репликации ColEl ori размером 589 п.н., имеющую координаты с 3937 по 4525 п.н.; энхансер и промотор цитомегаловируса размером 584 п.н., имеющий координаты с 36 по 619 п.н.; промотор РНК-полимеразы фага Т7 размером 19 п.н., имеющий координаты с 664 по 682 п.н.; фрагмент размером 1682 п. н., содержащий целевой ген Н5, имеющий координаты с 696 по 2377 п.н. Указанный технический результат достигается также тем, что получена стабилизированная форма белка гемагглютинина вируса гриппа A (H5N8), продуцируемая рекомбинантной плазмидной ДНК pVAX-H5 по п. 2 в организме животных и индуцирующий специфический иммунный ответ против высокопатогенного вируса гриппа A (H5N8), имеющий аминокислотную последовательность SEQ ID NO: 3 (фиг.10) длиной 551 а.к.о., включащий в себя четыре стабилизирующие замены в НА2 субъединице: H26/W26, К51/151, Е103Я103 и H106/R106 и тримеризующий домен фибритина бактериофага Т4, обеспечивающий формирование гомотримеров гемагглютинина. [4].
При иммунизации животных (мышей линии Balb/c) созданной ДНК-конструкцией регистрируется специфический гуморальный и Т-клеточный ответ.
Изобретение иллюстрируется следующими графическими материалами.
На фиг. 1 приведена карта плазмиды pVAX-H5. На фиг. 2 приведен электрофоретический анализ экспрессии гена h5 в трансфицированных клетках НЕК293 в 1% агарозном геле:
Дорожка 1 - маркеры молекулярной массы;
Дорожка 2 - продукт, полученный с помощью ОТ-ПЦР из тотальной РНК, выделенной с клеток НЕК293, трансфицированных pVAX-H5.
На фиг. 3 представлен анализ продукции белка НА/Н5 в клетках НЕК293 с помощью вестерн-блотинга:
Дорожка 1 - лизат нетрансфицированных клеток НЕК293;
Дорожка 2 - культуральная среда, собранная с нетрансфицированных клеток НЕК293;
Дорожка 3 - маркер молекулярных масс Precision Plus Protein Dual Color
Standards (Bio-rad, США).
Дорожка 4 - лизат клеток НЕК293, трансфицированных плазмидой pVAX-H5; Дорожка 5 - культуральная среда, собранная с клеток НЕК293,
трансфицированных плазмидой pVAX-H5.
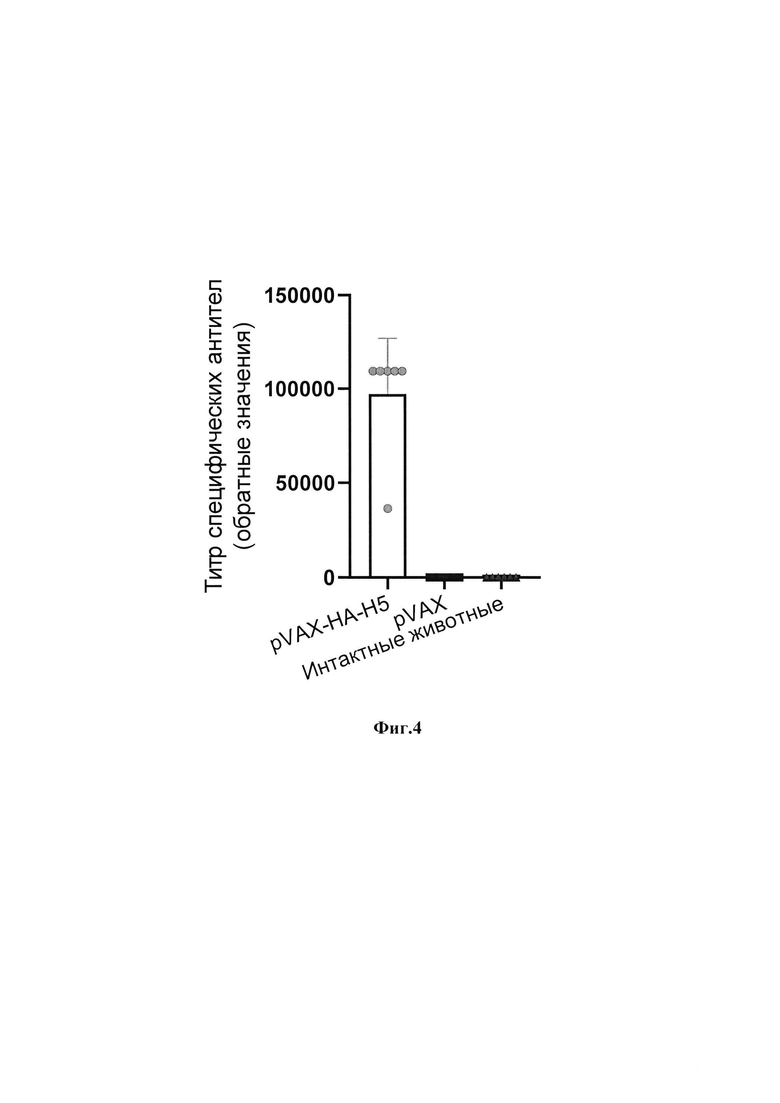
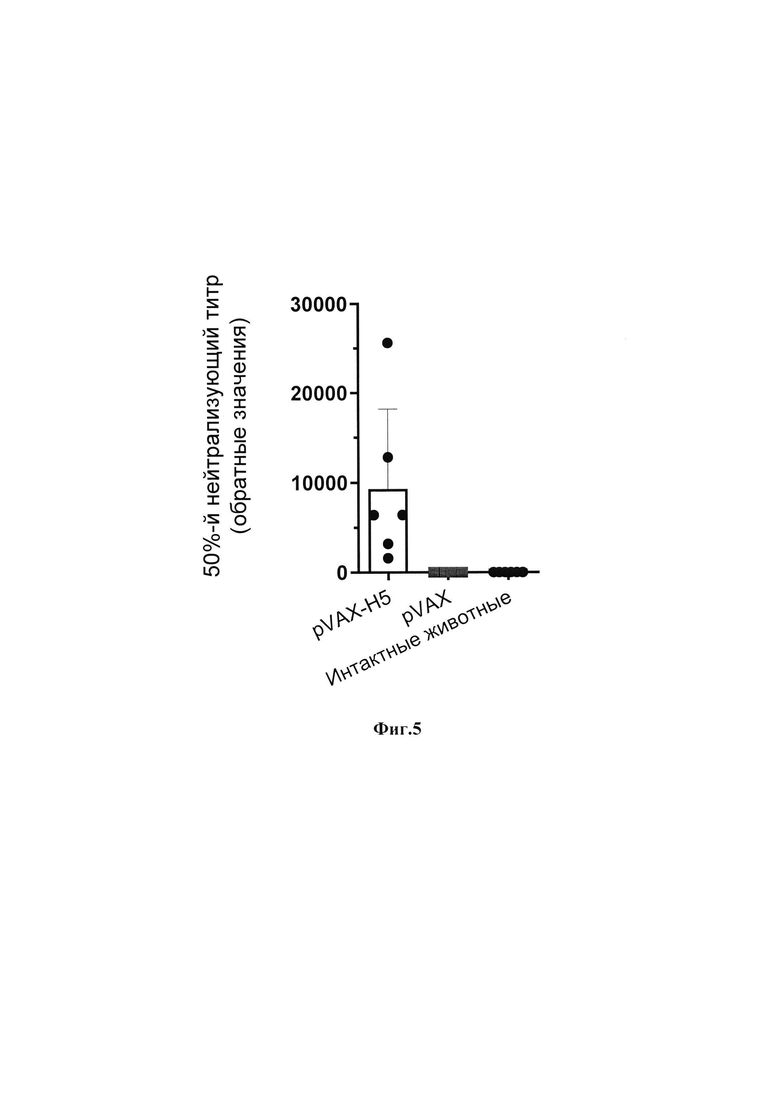
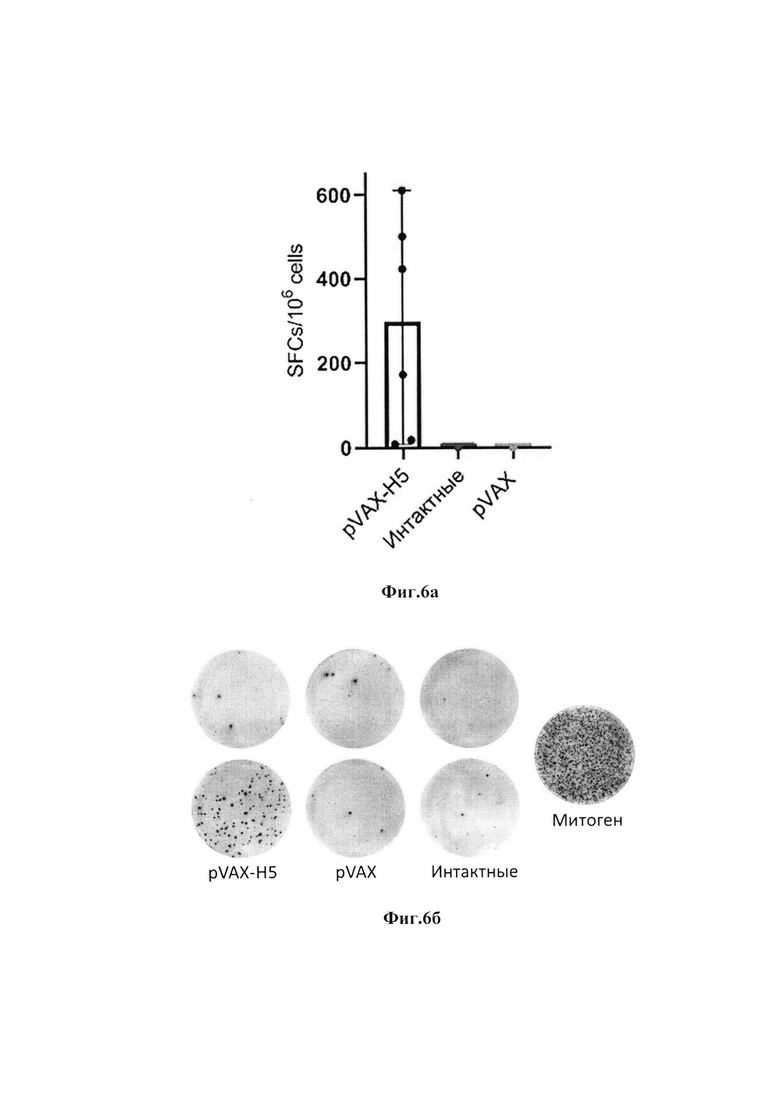
Дорожка 6 - рекомбинантный гемагглютинин НА/Н5 вируса гриппа A (H5N8) (К+). На фиг. 4 приведены титры антител в сыворотке иммунизированных животных. В качестве антигена для сорбции в ИФА был использован рекомбинантный белок Н5: pVAX-H5 - группа животных, иммунизированных ДНК-вакциной pVAX-H5; pVAX - группа животных, иммунизированных вектором pVAX; интактные животные - группа животных, не подвергавшиеся никаким манипуляциям. Данные представлены как среднее ± с.о. На фиг. 5 представлены данные вируснейтрализующей активности иммунных сывороток в отношении вируса гриппа A/turkey/Stavropol/320-01/2020 (H5N8). pVAX-H5 - группа животных, иммунизированных ДНК-вакциной pVAX-H5; pVAX - группа животных, иммунизированных вектором pVAX; Данные представлены как среднее ± с.о. На фиг. 6а приведены результаты оценки вирус-специфических Т-лимфоцитов у иммунизированных животных на 35 сутки после начала иммунизации с помощью метода ELISpot: pVAX-H5 -группа животных, иммунизированных ДНК-вакциной pVAX-H5; pVAX -группа животных, иммунизированных вектором pVAX; интактные животные - группа животных, не подвергавшиеся никаким манипуляциям. Данные представлены как среднее ± с.о. На фиг. 66 представлены репрезентативные изображения лунок планшетов после проведения анализа ELISpot. На фиг. 7 приведены кривые выживаемости, полученные в различных группах иммунизированных животных после заражения штаммом вируса гриппа A/Astrakhan/3212/2020 (H5N8): pVAX-H5 - группа животных, иммунизированных ДНК-вакциной pVAX-H5; pVAX - группа животных, иммунизированных вектором pVAX; интактные животные - группа животных, не подвергавшиеся никаким манипуляциям. Данные представлены как среднее ± с.о. На фиг. 8 приведена нуклеотидная последовательность (SEQ ID NO: 1) рекомбинантной плазмидной ДНК pVAX-H5. На фиг. 9 представлена нуклеотидная последовательность (SEQ ID NO: 2) гена h5, кодирующего стабилизированную форму белка гемагглютинина НА/Н5. На фиг. 10 приведена аминокислотная последовательность (SEQ ID NO: 3) белка гемагглютинина НА/Н5 вируса гриппа A (H5N8). Пример 1. Дизайн плазм иды pVAX-H5.
Для конструирования иммуногена использовали последовательность гена, кодирующего полноразмерный белок гемагглютинина Н5, источником которого стал вирус A/turkey/Stavropol/320-01/2020 (EPI_ISL_1114749). Для стабилизации гемагглютинина, в области рН-переключателей, уязвимой для протеаз, были внесены следующие замены: консервативные гистидины Н26 и HI06 были заменены на W (триптофан) и на R (аргинин), соответственно. Консервативные K51 и Е103 были заменены на алифатический изолейцин (151 и ПОЗ) [3]. Также для повышения стабилизации структуры был добавлен тримеризующий домен фибритина бактериофага Т4, обеспечивающий формирование гомотримеров гемагглютинина [4]. В результате была спроектирована аминокислотная последовательность SEQ ID N0:2 (фиг. 9) модифицированного полноразмерного гемагглютинина вируса гриппа A (H5N8), обозначенного как НА/Н5. Ген h5, кодирующий НА/Н5, синтезировали в ООО «ДНК-синтез» (Россия) и встраивали в вектор pVAX1 ("Thermo Fisher Scientific", США) по сайтам AsuNHI и CciNI. В результате была получена ДНК-конструкция, кодирующая НА/Н5, которая была обозначена как pVAX-H5 (фиг. 1), имеющая нуклеотидную последовательность SEQ ID NO: 1 (фиг. 8).
Пример 2. Наработка и очистка рекомбинантной плазмиды pVAX-H5. Клетки Escherichia coli Stbl3 трансформировали плазмидой pVAX-H5 с помощью хлористого кальция. Наработку плазмидной ДНК для иммунизации проводили в 2.7 л питательной среды LB с добавлением канамицина (25 мкг/мл). Плазмидную ДНК выделяли и очищали с помощью набора EndoFree Plasmid Giga Kit (QIAGEN, Германия) согласно рекомендациям производителя. Данный набор позволяет эффективно очистить препарат плазмидной ДНК от бактериальных эндотоксинов. После очистки структуру плазмиды подтвердили рестрикционным анализом, концентрацию и чистоту - спектрофотометрией.
Пример 3. Проверка эффективности экспрессии целевого гена в условиях in vitro.
3.1 Трансфекция клеток НЕК293 Перевиваемые клетки почечного эмбрионального эпителия человека НЕК293 (Human embryonic kidney) были получены из коллекции клеточных культур ГНЦ ВБ «Вектор». Культуру клеток НЕК293 культивировали при температуре 37°С, 70% относительной влажности воздуха и 5% содержании СО2. В качестве ростовой среды использовали среду DMEM с добавлением 10% термоинактивированной (30 мин при температуре 56°С) фетальной бычьей сыворотки (FBS), 2 мМ L-глутамина и антибиотика (гентамицин, 50 мкг/мл). Трансфекцию клеток плазмидой pVAX-H5 проводили с использованием липофектамина (Lipofectamin 3000) по методике, рекомендованной фирмой-производителем. По истечении 48 часов после трансфекции собирали культуральную среду, а клетки снимали с использованием раствора Версена и центрифугировали 5000 об/мин, осадок растворяли в 30 мкл PBS для дальнейшей работы.
3.2 Исследование экспрессии генов на уровне мРНК
Для проведения ОТ-ПЦР из трансфицированных клеток выделяли тотальную РНК с использованием реагента «ЛИРА» для выделения РНК, ДНК и белков (Биолабмикс, Россия) согласно рекомендации производителя.
Анализ экспрессии гена был проведен с помощью обратной транскрипции с ПЦР в одной пробирке набором БиоМастер ОТ-ПЦР-Экстра (2*) (Биолабмикс, Россия). Реакцию обратной транскрипции проводили в общем объеме 50 мкл: 2х смесь для ОТ-ПЦР-Экстра - 25 мкл, прямой праймер (5'-TTTCTGGCTAGCGCCGCCACCATGGAGAACA-3/) с концентрацией 10 пМ -2,5 мкл, обратный праймер (5'-AAAAAAAGCGGCCGCTCATTACAGGAAGGT-3') с концентрацией 10 пМ - 2,5 мкл, РНК-матрицу - 5 мкл, БиоМастер Экстра-Микс - 2 мкл, и стерильная вода до общего объема 50 мкл. Готовую реакционную смесь перемещали в амплификатор (Veriti, Thermo Fisher Scientific, США). ОТ-ПЦР проводили, используя следующий режим - обратная транскрипция - 55°С в течение 30 минут, предварительная денатурация - 93°С в течение 5 минут, затем 35 циклов: 93°С - 15 секунд, 64°С - 15 секунд, 68°С - 45 секунд, финальная элонгация при 68°С в течение 7 минут. После проведения ОТ-ПЦР продукты амплификации анализировали с помощью электрофореза в 1%-м агарозном геле. На электрофореграмме (фиг. 2) видно, что размер амплифицированного фрагмента составляет примерно 1600 п. о., что соответствует теоретически рассчитанному фрагменту при использовании указанных специфических праймеров.
3.3 Исследование продукции целевого белка
Лизат трансфицированных клеток и культуральную среду, в которой они росли, исследовали на наличие целевого белка с помощью иммуноблотинга с использованием положительной сыворотки хорька, зараженного вирусом гриппа A (H5N8). Было показано наличие целевого белкового продукта в культуральной среде, собранной с клеток, трансфицированных плазмидой pVAX-H5, и в их лизате (фиг. 3).
Пример 4. Иммуногенность ДНК-вакцинной конструкции pVAX-H5 4.1 Иммунизация лабораторных животных, подготовка образцов для исследования
Работа с животными проводилась в соответствии с «Руководством по уходу и использованию лабораторных животных». Протоколы были одобрены Комитетом по уходу и использованию животных (IACUC) при Государственном научном центре вирусологии и биотехнологии «Вектор» (Протокол БЭК №1 от 21.03.2023).
Для оценки иммуногенности созданной конструкции использовали самок мышей BALB/c массой 16-18 г. Мышей разделили на три группы по 6 животных в каждой. 1-й группе вводили 100 мкг плазмиды pVAX-H5 в 50 мкл физиологического раствора; 2-й группе вводили 100 мкг плазмиды pVAX в 50 мкл физиологического раствора; 3-я группа состояла из интактных животных. Мышей иммунизировали дважды внутримышечно в дни 0 и 21 с помощью струйной инжекции.
Струйную инжекцию проводили с помощью инжектора HYALURON PEN XY PLUS (Китай) с использованием индивидуальных насадок-сопло. Параметры введения препарата составляли: скорость струи 220 м/с, давление 6,5 бар, время инжекции 0,33 с. Для снижения болевого синдрома применяли ингаляционный наркоз животных (RWD) с 2,5% изофлурана. При иммунизации животных сначала помещали в индукционную камеру наркозной системы, в которую подается анестезирующий газ для введения в наркоз, а затем животных перемещали под наркозные маски для осуществления дальнейших манипуляций.
Волосяной покров удаляли с бедра с помощью геля для депиляции, далее проводили процедуру инжекции.
Через две недели после второй иммунизации у животных брали кровь для анализа гуморального иммунного ответа и собирали селезенки для анализа клеточного ответа. Сыворотку отделяли от клеточных элементов центрифугированием (9000 g, 15 мин), нагревали в течение 30 мин при 56°С и исследовали на наличие антител, специфически связывающихся с белком Н5, в ИФА. Образцы также были проанализированы на их нейтрализующую активность. Селезенки последовательно измельчали на нейлоновых фильтрах для клеток с диаметром пор 70 и 40 мкм (Jet Biofil, Китай). После лизиса эритроцитов лизирующим буфером (Sigma, США) спленоциты дважды промывали полной средой RPMI и помещали в 1 мл среды RPMI с 2 мМ L-глутамина, 50 мкг/мл гентамицина и 10% FBS (Thermo Fisher Scientific, США). Клетки подсчитывали с использованием автоматического счетчика клеток ТС20 (Bio-Rad, США). Среди спленоцитов количество IFN-y-продуцирующих клеток определяли методом ELISpot.
4.2 Иммуноферментный анализ сывороток, исследование их вируснейтрализующих свойств
В качестве антигена для ИФА использовали рекомбинантный эукариотический белок НА/Н5, сорбированный на 96-луночные планшеты (Corning, США) в концентрации 1 мкг/мл в 2 М мочевине в течение ночи при 4°С. Планшеты трижды промывали и инкубировали в течение 1,5 ч при комнатной температуре с 1% казеина в PBS с 0,05% Tween 20 для блокирования неспецифического связывания белка. Затем планшеты инкубировали с трехкратными серийными разведениями мышиной сыворотки в течение 1 ч при комнатной температуре, после промывки PBST добавляли кроличьи антимышиные IgG, конъюгированные с пероксидазой хрена (Sigma, США), разведенные 1:3000, и инкубировали 1 ч при комнатной температуре. После окончательной промывки планшеты проявляли с использованием субстрата ТМВ (Amresco, США). Реакцию останавливали IN НС1 и анализировали при 450 нм на многорежимном ридере для микропланшетов Varioskan LUX (Thermo Fisher Scientific, США).
По результатам ИФА в конечной точке эксперимента титры НА/Н5-специфических антител были выявлены только в группе животных, иммунизированных pVAX-H5, средний титр составил 1:100000 (фиг. 4).
Нейтрализующие свойства сывороток крови определяли по ингибированию цитопатического действия (ЦПД) вируса в культуре клеток in vitro [5]. Через двое суток после начала инкубации клеток MDCK со смесью серийных разведений сывороток и 100 ТСГО50/лунка штамма A/turkey/Stavropol/320-01/2020 клетки окрашивали раствором кристаллического фиолетового (1,3 г красителя растворяли в 50 мл 96% этилового спирта, доводили дистиллированной водой до 700 мл и добавляли 300 мл 40% раствора формалина) и анализировали с помощью многорежимного ридера для визуализации клеток Agilent BioTek Cytation 5 (Thermo fisher scientific). За титр нейтрализации принимали значение самого высокого разведения сыворотки, при котором достигается>50% нейтрализация вируса, что соответствует>50% жизнеспособных клеток.
В результате было установлено, что сыворотки животных, иммунизированных ДНК-вакциной pVAX-H5, способны нейтрализовать живой вирус гриппа штамм A/turkey/Stavropol/320-01/2020 со средним титром 1:10000 (фиг. 5). Нейтрализующей активности контрольных сывороток обнаружено не было.
4.3 Исследование поствакцинального Т-клеточного ответа с помощью ELISpot
Анализ Т-клеточного иммунного ответа проводили с использованием набора Mouse IFN-y ELISpot HRP (Mabtech, Швеция) в соответствии с инструкциями производителя. Спленоциты пассировали в количестве 2,5*105 клеток/лунку и добавляли среду RPMI с 10% FBS (отрицательный контроль), или смесь пептидов (каждого в концентрации 20 мкг/мл), или конканавалин А (положительный контроль). Клетки инкубировали в течение 20 ч при 37°С в присутствии 5% СО2. Затем планшеты промывали и инкубировали с первичными антителами против IFN-y. Планшеты снова промывали и затем инкубировали со вторичным антителом, конъюгированным со пероксидазой хрена. После промывки планшеты инкубировали с ТМВ. С помощью ELISpot было показано, что только в группе, иммунизированной ДНК-вакциной, активируется специфический клеточный иммунитет, выраженный в значительном повышении количества лимфоцитов, продуцирующих IFN-y (фиг. 66). Пептиды были подобраны в соответствии с эпитопами, входящими в состав иммуногена и предсказанными с использованием инструментов IEDB Analysis Resource и синтезированы AtaGenix Laboratories (Ухань, Китай); чистота пептидов >80% (табл. 1).
В группе, иммунизированной pVAX-H5, среднее количество Т-лимфоцитов, секретирующих IFN-y, в расчете на 106 спленоцитов составило 300 (фиг. 6а). Значения в опытной группе достоверно отличались от контрольных групп.
5. Исследование способности прототипа ДНК-вакцины защищать от заражения вирусом A/turkey/Stavropol/320-01/2020 (H5N8)
Анализ способности прототипа ДНК-вакцины защищать от заражения вирусом был проведен с использованием мышей линии BALB/c, которые являются чувствительными к заражению вирусом гриппа A/Astrakhan/3212/2020 (H5N8). Для анализа были сформированы следующие группы по 10 мышей в каждой: 1-й группе вводили 100 мкг pVAX-H5; 2-й группе вводили 100 мкг pVAX; 3-й группе вводили инактивированный вирус гриппа A (H5N8) внутримышечно в комплексе с неполным адъювантом Фрейда в качестве положительного контроля; 4-я группа состояла из интактных животных и служила отрицательным контролем. Иммунизацию проводили двухкратно с интервалом в 21 день. Через 14 дней после второй иммунизации, мышей заражали интраназально дозой 20 МЛД50 штаммом вируса гриппа A/Astrakhan/3212/2020 (H5N8), гомологичного вакцинному штамму. Манипуляции по заражению мышей проводили на фоне наркоза смесью препаратов ЗолетилЮО и Ксила. Наблюдение за животными после заражения осуществлялось ежедневно в течении 14 дней после заражения, одновременно вели наблюдение за клиническими признаками в качестве индикатора заболевания (активность, аппетит) и гибелью.
По итогам проведенного эксперимента установлено, что протективность в группах мышей, иммунизированных ДНК-вакциной pVAX-H5 и инактивированным вирусом, составила 100%. В группе, получавшей исходный вектор pVAX, погибло 30%, при этом следует отметить, что в данной группе у всех животных проявлялись признаки заболевания - снижение активности и аппетита, чего не наблюдалось в группах мышей, которым вводили прототипы ДНК-вакцин (фиг. 7). Частичная гибель в группе pVAX может быть связана с одной стороны с недостаточной дозой вируса (20 МЛД50 обеспечивает 95-99% гибели), с другой стороны, известно, что плазмидная ДНК обладает адъювантными свойствами и активируют систему врожденного иммунитета [6].
Источники патентной и научно-технической информации.
1. Jiang Y. et al. Enhanced protective efficacy of H5 subtype avian influenza DNA vaccine with codon optimized HA gene in a pCAGGS plasmid vector // Antiviral research. - 2007. - T. 75. - №. 3. - C. 234-241.
2. Kodihalli S., Kobasa D.L., Webster R.G. Strategies for inducing protection against avian influenza A virus subtypes with DNA vaccines // Vaccine. - 2000. - T. 18.-№. 23. - C. 2592-2599.
3. Milder F. J. et al. Universal stabilization of the influenza hemagglutinin by structure-based redesign of the pH switch regions // Proceedings of the National Academy of Sciences. - 2022. - Т. 119. - №. 6. - C. e2115379119.
4. Lu, Y.; Welsh, J.P.; Swartz, J.R. Production and stabilization of the trimeric influenza hemagglutinin stem domain for potentially broadly protective influenza vaccines. Proceedings of the National Academy of Sciences 2014, 111, 125-130. doi: 10.1073/pnas.l308701110.
5. Gross FL, Bai Y, Jefferson S, Holiday C, Levine MZ. Measuring Influenza Neutralizing Antibody Responses to A(H3N2) Viruses in Human Sera by Microneutralization Assays Using MDCK-SIAT1 Cells // J Vis Exp.- 2017. - T. 129.
6. Martin J. E. et al. A DNA vaccine for Ebola virus is safe and immunogenic in a phase I clinical trial // Clinical and vaccine immunology. - 2006. - T. 13. - №. 11. -C. 1267-1277.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing originalFreeTextLanguageCode="ru"
nonEnglishFreeTextLanguageCode="ru" dtdVersion="V1_3"
fileName="pVAX_H5_02022024.xml" softwareName="WIPO Sequence"
softwareVersion="2.3.0" productionDate="2024-10-31">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>1234567</ApplicationNumberText>
<FilingDate>2024-02-02</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>123456</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>12345678</ApplicationNumberText>
<FilingDate>2024-01-31</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">Федеральное бюджетное учреждение
науки «Государственный научный центр вирусологии и биотехнологии
«Вектор» Федеральной службы по надзору в сфере защиты прав
потребителей и благополучия человека (ФБУН ГНЦ ВБ «Вектор»
Роспотребнадзора)</ApplicantName>
<ApplicantNameLatin>Federalnoe byudzhetnoe uchrezhdenie nauki
"Gosudarstvennyj nauchnyj tsentr virusologii i biotekhnologii
"Vektor" Federalnoj sluzhby po nadzoru v sfere zashchity
prav potrebitelej i blagopoluchiya cheloveka (FBUN GNTS VB
"Vektor" Rospotrebnadzora) (RU)</ApplicantNameLatin>
<InventionTitle languageCode="ru">Ген h5, кодирующий
стабилизированную форму белка гемагглютинина HA/H5 вируса гриппа А
(H5N8), рекомбинантная плазмидная ДНК pVAX-H5, содержащая ген h5 и
обеспечивающая его экспрессию и белок гемагглютинин HA/H5,
продуцируемый рекомбинантной плазмидной ДНК pVAX-H5 в организме
животных и индуцирующий специфический иммунный ответ против
высокопатогенного вируса гриппа А (H5N8)</InventionTitle>
<SequenceTotalQuantity>3</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>4590</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..4590</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gctgcttcgcgatgtacgggccagatatacgcgttgacattgattattg
actagttattaatagtaatcaattacggggtcattagttcatagcccatatatggagttccgcgttacat
aacttacggtaaatggcccgcctggctgaccgcccaacgacccccgcccattgacgtcaataatgacgta
tgttcccatagtaacgccaatagggactttccattgacgtcaatgggtggactatttacggtaaactgcc
cacttggcagtacatcaagtgtatcatatgccaagtacgccccctattgacgtcaatgacggtaaatggc
ccgcctggcattatgcccagtacatgaccttatgggactttcctacttggcagtacatctacgtattagt
catcgctattaccatggtgatgcggttttggcagtacatcaatgggcgtggatagcggtttgactcacgg
ggatttccaagtctccaccccattgacgtcaatgggagtttgttttggcaccaaaatcaacgggactttc
caaaatgtcgtaacaactccgccccattgacgcaaatgggcggtaggcgtgtacggtgggaggtctatat
aagcagagctctctggctaactagagaacccactgcttactggcttatcgaaattaatacgactcactat
agggagacccaagctggctagcgccgccaccatggagaacatcgtgctgctgctggccatcgtgagcctg
gtgaagagcgaccagatctgcatcggctaccacgccaacaacagcaccgagcaggtggacaccatcatgg
agaagaacgtgaccgtgacccacgcccaggacatcctggagaagacccacaacggcaagctgtgcgacct
gaacggcgtgaagcccctgatcctgaaggactgcagcgtggccggctggctgctgggcaaccccatgtgc
gacgagttcatcagggtgcccgagtggagctacatcgtggagagggccaaccccgccaacgacctgtgct
accccggcagcctgaacgactacgaggagctgaagcacctgctgagcaggatcaaccacttcgagaagat
cctgatcatccccaagagcagctggcccaaccacgagaccagcctgggcgtgagcgccgcctgcccctac
cagggcgcccccagcttcttcaggaacgtggtgtggctgatcaagaagaacgacgcctaccccaccatca
agatcagctacaacaacaccaacagggaggacctgctgatcctgtggggcatccaccacagcaacaacgc
cgaggagcagaccaacctgtacaagaaccccaccacctacatcagcgtgggcaccagcaccctgaaccag
aggctggtgcccaagatcgccaccaggagccaggtgaacggccagaggggcaggatggacttcttctgga
ccatcctgaagcccgacgacgccatccacttcgagagcaacggcaacttcatcgcccccgagtacgccta
caagatcgtgaagaagggcgacagcaccatcatgaagagcggcgtggagtacggccactgcaacaccaag
tgccagacccccgtgggcgccatcaacagcagcatgcccttccacaacatccaccccctgaccatcggcg
agtgccccaagtacgtgaagagcaacaagctggtgctggccaccggcctgaggaacagccccctgaggga
gaagcagaccaggggcctgttcggcgccatcgccggcttcatcgagggcggctggcagggcatggtggac
ggctggtacggctacaggtggagcaacgagcagggcagcggctacgccgccgacaaggagagcacccaga
aggccatcgacggcgtgaccaacatcgtgaacagcatcatcgacaagatgaacacccagttcgaggccgt
gggcagggagttcaacaacctggagaggaggatcgagaacctgaacaagaagatggaggacggcttcctg
gacgtgtggacctacaacgccgagctgctggtgctgatgatcaacgagaggaccctggacttccacgaca
gcaacgtgaagaacctgtacgacaaggtgaggctgcagctgagggacaacgccaaggagctgggcaacgg
ctgcttcgagttctaccacaagtgcgacaacgagtgcatggagagcgtgaggaacggcacctacgactac
ccccagtacagcgaggaggccaggctgaagagggaggagatcagcggcgtgaagggcagcggcggctaca
tccccgaggcccccagggacggccaggcctacgtgaggaaggacggcgagtgggtgctgctgagcacctt
cctgtaatgagcggccgctcgagtctagagggcccgtttaaacccgctgatcagcctcgactgtgccttc
tagttgccagccatctgttgtttgcccctcccccgtgccttccttgaccctggaaggtgccactcccact
gtcctttcctaataaaatgaggaaattgcatcgcattgtctgagtaggtgtcattctattctggggggtg
gggtggggcaggacagcaagggggaggattgggaagacaatagcaggcatgctggggatgcggtgggctc
tatggcttctactgggcggttttatggacagcaagcgaaccggaattgccagctggggcgccctctggta
aggttgggaagccctgcaaagtaaactggatggctttctcgccgccaaggatctgatggcgcaggggatc
aagctctgatcaagagacaggatgaggatcgtttcgcatgattgaacaagatggattgcacgcaggttct
ccggccgcttgggtggagaggctattcggctatgactgggcacaacagacaatcggctgctctgatgccg
ccgtgttccggctgtcagcgcaggggcgcccggttctttttgtcaagaccgacctgtccggtgccctgaa
tgaactgcaagacgaggcagcgcggctatcgtggctggccacgacgggcgttccttgcgcagctgtgctc
gacgttgtcactgaagcgggaagggactggctgctattgggcgaagtgccggggcaggatctcctgtcat
ctcaccttgctcctgccgagaaagtatccatcatggctgatgcaatgcggcggctgcatacgcttgatcc
ggctacctgcccattcgaccaccaagcgaaacatcgcatcgagcgagcacgtactcggatggaagccggt
cttgtcgatcaggatgatctggacgaagagcatcaggggctcgcgccagccgaactgttcgccaggctca
aggcgagcatgcccgacggcgaggatctcgtcgtgacccatggcgatgcctgcttgccgaatatcatggt
ggaaaatggccgcttttctggattcatcgactgtggccggctgggtgtggcggaccgctatcaggacata
gcgttggctacccgtgatattgctgaagagcttggcggcgaatgggctgaccgcttcctcgtgctttacg
gtatcgccgctcccgattcgcagcgcatcgccttctatcgccttcttgacgagttcttctgaattattaa
cgcttacaatttcctgatgcggtattttctccttacgcatctgtgcggtatttcacaccgcatacaggtg
gcacttttcggggaaatgtgcgcggaacccctatttgtttatttttctaaatacattcaaatatgtatcc
gctcatgagacaataaccctgataaatgcttcaataatagcacgtgctaaaacttcatttttaatttaaa
aggatctaggtgaagatcctttttgataatctcatgaccaaaatcccttaacgtgagttttcgttccact
gagcgtcagaccccgtagaaaagatcaaaggatcttcttgagatcctttttttctgcgcgtaatctgctg
cttgcaaacaaaaaaaccaccgctaccagcggtggtttgtttgccggatcaagagctaccaactcttttt
ccgaaggtaactggcttcagcagagcgcagataccaaatactgtccttctagtgtagccgtagttaggcc
accacttcaagaactctgtagcaccgcctacatacctcgctctgctaatcctgttaccagtggctgctgc
cagtggcgataagtcgtgtcttaccgggttggactcaagacgatagttaccggataaggcgcagcggtcg
ggctgaacggggggttcgtgcacacagcccagcttggagcgaacgacctacaccgaactgagatacctac
agcgtgagctatgagaaagcgccacgcttcccgaagggagaaaggcggacaggtatccggtaagcggcag
ggtcggaacaggagagcgcacgagggagcttccagggggaaacgcctggtatctttatagtcctgtcggg
tttcgccacctctgacttgagcgtcgatttttgtgatgctcgtcaggggggcggagcctatggaaaaacg
ccagcaacgcggcctttttacggttcctggccttttgctggccttttgctcacatgttctt</INSDSeq
_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>1682</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..1682</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q7">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gctagcgccgccaccatggagaacatcgtgctgctgctggccatcgtga
gcctggtgaagagcgaccagatctgcatcggctaccacgccaacaacagcaccgagcaggtggacaccat
catggagaagaacgtgaccgtgacccacgcccaggacatcctggagaagacccacaacggcaagctgtgc
gacctgaacggcgtgaagcccctgatcctgaaggactgcagcgtggccggctggctgctgggcaacccca
tgtgcgacgagttcatcagggtgcccgagtggagctacatcgtggagagggccaaccccgccaacgacct
gtgctaccccggcagcctgaacgactacgaggagctgaagcacctgctgagcaggatcaaccacttcgag
aagatcctgatcatccccaagagcagctggcccaaccacgagaccagcctgggcgtgagcgccgcctgcc
cctaccagggcgcccccagcttcttcaggaacgtggtgtggctgatcaagaagaacgacgcctaccccac
catcaagatcagctacaacaacaccaacagggaggacctgctgatcctgtggggcatccaccacagcaac
aacgccgaggagcagaccaacctgtacaagaaccccaccacctacatcagcgtgggcaccagcaccctga
accagaggctggtgcccaagatcgccaccaggagccaggtgaacggccagaggggcaggatggacttctt
ctggaccatcctgaagcccgacgacgccatccacttcgagagcaacggcaacttcatcgcccccgagtac
gcctacaagatcgtgaagaagggcgacagcaccatcatgaagagcggcgtggagtacggccactgcaaca
ccaagtgccagacccccgtgggcgccatcaacagcagcatgcccttccacaacatccaccccctgaccat
cggcgagtgccccaagtacgtgaagagcaacaagctggtgctggccaccggcctgaggaacagccccctg
agggagaagcagaccaggggcctgttcggcgccatcgccggcttcatcgagggcggctggcagggcatgg
tggacggctggtacggctacaggtggagcaacgagcagggcagcggctacgccgccgacaaggagagcac
ccagaaggccatcgacggcgtgaccaacatcgtgaacagcatcatcgacaagatgaacacccagttcgag
gccgtgggcagggagttcaacaacctggagaggaggatcgagaacctgaacaagaagatggaggacggct
tcctggacgtgtggacctacaacgccgagctgctggtgctgatgatcaacgagaggaccctggacttcca
cgacagcaacgtgaagaacctgtacgacaaggtgaggctgcagctgagggacaacgccaaggagctgggc
aacggctgcttcgagttctaccacaagtgcgacaacgagtgcatggagagcgtgaggaacggcacctacg
actacccccagtacagcgaggaggccaggctgaagagggaggagatcagcggcgtgaagggcagcggcgg
ctacatccccgaggcccccagggacggccaggcctacgtgaggaaggacggcgagtgggtgctgctgagc
accttcctgtaatgagcggccgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>551</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..551</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MENIVLLLAIVSLVKSDQICIGYHANNSTEQVDTIMEKNVTVTHAQDIL
EKTHNGKLCDLNGVKPLILKDCSVAGWLLGNPMCDEFIRVPEWSYIVERANPANDLCYPGSLNDYEELKH
LLSRINHFEKILIIPKSSWPNHETSLGVSAACPYQGAPSFFRNVVWLIKKNDAYPTIKISYNNTNREDLL
ILWGIHHSNNAEEQTNLYKNPTTYISVGTSTLNQRLVPKIATRSQVNGQRGRMDFFWTILKPDDAIHFES
NGNFIAPEYAYKIVKKGDSTIMKSGVEYGHCNTKCQTPVGAINSSMPFHNIHPLTIGECPKYVKSNKLVL
ATGLRNSPLREKQTRGLFGAIAGFIEGGWQGMVDGWYGYRWSNEQGSGYAADKESTQKAIDGVTNIVNSI
IDKMNTQFEAVGREFNNLERRIENLNKKMEDGFLDVWTYNAELLVLMINERTLDFHDSNVKNLYDKVRLQ
LRDNAKELGNGCFEFYHKCDNECMESVRNGTYDYPQYSEEARLKREEISGVKGSGGYIPEAPRDGQAYVR
KDGEWVLLSTFL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---



Изобретение относится к области биотехнологии, конкретно к рекомбинантному получению вирусных антигенных белков, и может быть использовано для получения белка гемагглютинина НА/Н5 вируса гриппа A (H5N8). Предложены ген H5, кодирующий стабилизированную форму белка гемагглютинина НА/Н5 вируса гриппа A (H5N8), рекомбинантная плазмидная ДНК pVAX-H5, содержащая указанный ген H5, и белок гемагглютинина НА/Н5, продуцируемый рекомбинантной плазмидной ДНК pVAX-H5 в клетках эукариот (организме животных) и индуцирующий специфический иммунный ответ против высокопатогенного вируса гриппа А (H5N8). Изобретение позволяет создать ДНК-вакцину, способную индуцировать НА/Н5-специфический гуморальный и клеточный ответ для защиты от заболевания, вызванного вирусом гриппа A (H5N8). 3 н.п. ф-лы, 10 ил., 1 табл., 4 пр.
1. Ген Н5, кодирующий стабилизированную форму белка гемагглютинина вируса гриппа A (H5N8), имеющий последовательность SEQ ID N0: 2 размером 1682 п.н. и содержащий на 5'-конце последовательность Козак GCCGCCACCATGGAG и сайт для эндонуклеазы рестрикции AsuNHI, а на 3'-конце - два стоп-кодона ТАА и TGA и сайт для эндонуклеазы рестрикции CciNI.
2. Рекомбинантная плазмидная ДНК pVAX-H5, направленная на продукцию стабилизированной формы белка гемагглютинина вируса гриппа A (H5N8), имеющая нуклеотидную последовательность SEQ ID NO: 1 размером 4590 п.н., молекулярный вес 2838763 Да, содержащая целевой ген Н5 по п. 1, кодирующий стабилизированную форму указанного белка гемагглютинина, перед которым находится энхансер и промотор CMV, обеспечивающий транскрипцию гена в эукариотической системе, и состоящая из следующих фрагментов:
- векторный фрагмент 2367-710 плазмиды pVAX размером 2934 п.н., содержащий bGH поли(А) сигнал и имеющий координаты с 2420 по 2644 п.н.; ген устойчивости к неомицину/канамицину NeoR/KanR размером 795 п.н., имеющий координаты с 2817 по 3611 п.н.; точку начала репликации ColEl ori размером 589 п.н., имеющую координаты с 3937 по 4525 п.н.; энхансер и промотор цитомегаловируса размером 584 п.н., имеющий координаты с 36 по 619 п.н.; промотор РНК-полимеразы фага Т7 размером 19 п.н., имеющий координаты с 664 по 682 п.н.;
- фрагмент размером 1682 п.н., содержащий целевой ген Н5, имеющий координаты с 696 по 2377 п.н.
3. Стабилизированная форма белка гемагглютинина вируса гриппа А (H5N8), продуцируемая рекомбинантной плазмидной ДНК pVAX-H5 по п. 2 в организме животных и индуцирующая специфический иммунный ответ против высокопатогенного вируса гриппа A (H5N8), имеющий аминокислотную последовательность SEQ ID NO: 3 длиной 551 а.к.о., включащий в себя четыре стабилизирующие замены в НА2 субъединице: H26/W26, К51/151, Е103/1103 и H106/R106 и тримеризующий домен фибритина бактериофага Т4, обеспечивающий формирование гомотримеров гемагглютинина.
| CN 106039303 A, 26.10.2016, MILDER F | |||
| J | |||
| Способ получения продуктов конденсации фенолов с формальдегидом | 1924 |
|
SU2022A1 |
Авторы
Даты
2024-12-24—Публикация
2024-03-11—Подача