Изобретение относится к плазмидной ДНК pBSI-COV-Ub, обеспечивающей экспрессию гена bsi-cov-ub, кодирующего искусственный иммуноген вируса SARS-CoV-2 в клетках эукариот, и может быть использовано в области биотехнологии, медицины и иммунологии. Плазмида pBSI-COV-Ub индуцирует SARS-CoV-2-специфический Т-клеточный ответ у лабораторных животных.
Существует ряд подходов к созданию вакцин, и многие из них были реализованы во время борьбы с пандемией COVID-19. Изучение поствакцинального иммунного ответа привело к пониманию огромной важности клеточного звена иммунитета в формировании защиты от заболевания COVID-19. Было показано, что вирус-специфический клеточный ответ является более пролонгированным и более консервативным, что очень важно для таких быстро меняющих антигенный спектр вирусов, как SARS-CoV-2 [1]. Вакцины против SARS-CoV-2 на основе мРНК и векторные вакцины на основе аденовирусов (Спутник-V), кодирующие ген S-белка коронавируса, способны индуцировать высокий уровень анти-SARS-CoV-2 антител [2-6], однако через 6 месяцев титр антител падает в 10 и более раз, в результате требуются повторные вакцинации для формирования защитного уровня антител. Известно, что для формирования продолжительно и стойкого противовирусного иммунного ответа необходимо активировать помимо гуморального, также и клеточный иммунный ответ.
Недавние исследования показали, что люди, вакцинированные мРНК-вакциной, имели умеренный уровень ответа CD8+Т-клеток на эпитопы спайкового (S) белка [7], а Т-клеточный ответ на эпитопы других белков вируса SARS-CoV-2 отсутствовал. У лиц, перенесших инфекцию и выздоровевших, определяется более широкий спектр CD8+, специфичным эпитопам разных белков SARS-CoV-2. Этот факт заставляет обратить внимание исследователей на разработку Т-клеточных иммуногенов, обеспечивающих широкую защиту.
При проектировании Т-клеточных вакцин исследователи часто включают в состав иммуногена последовательность убиквитина. В ряде исследований было показано, что присоединение убиквитина к белковому иммуногену способствует нацеливанию его на протеасому, что приводит к эффективному процессингу, высвобождению пептидов-эпитопов, которые презентируются MHC I на поверхности антигенпрезентирующей клетки и способствуют активации цитотоксических (CD8+) Т-лимфоцитов и (CD4+) Т-хелперов [8-10]. Т.о., повышается вирус-специфический клеточный иммунный ответ, опосредованный CD4+и CD8+Т-лимфоцитами.
Для доставки Т-клеточного иммуногена можно использовать ДНК-вакцины, мРНК-вакцины и вирусные вектора. ДНК-вакцины обладают рядом преимуществ по сравнению с другими платформами: в отличие от мРНК-вакцин они гораздо более термостабильны; в отличие от векторных вакцин они не основаны на инфекционном агенте, против плазмиды-носителя не формируется иммунитет. ДНК-вакцины легко нарабатываются в больших количествах и способны обеспечить индукцию Т-клеточного иммунитета за счет обеспечения синтеза целевого белка внутри клетки, что приводит к естественной презентации фрагментов эндогенных белков молекулами MHC I и II класса.
Наиболее близким аналогом (прототипом) является ДНК-вакцина ZyCoV-D, разработанная и произведенная индийской компанией Cadila Healthcare Ltd, (международная заявка WO2021214703, МПК A61K 39/215, опубл. 28.10.2021 г.), одобренная для массового применения в Индии в целях иммунопрофилактики COVID-19 в экстренных условиях. Данная вакцина состоит из векторной плазмиды pVAX1, несущей ген S-белка вируса SARS-CoV-2 с сигнальным пептидом из IgE, и вводится внутрикожно с помощью безыгольного струйного инжектора PharmaJet Tropis. На животных моделях показана безопасность и эффективность данной вакцины: отсутствие встраивания рекомбинантной ДНК в геном, индукция обоих звеньев иммунитета [11, 12]. Однако стоит отметить и то, что иммунный ответ, направленный только на поверхностный белок вируса, со временем становится более слабым в результате множественных мутационных изменений, происходящих в результате эволюции вируса во время персистенции его в популяции. Поэтому использование в составе ДНК-вакцины ZyCoV-D только S-белка не может обеспечить длительного эффективного клеточного ответа, и необходимо включать в состав ДНК-вакцин эпитопы из других белков коронавируса [13]. У людей, переболевших родственным вирусом SARS-CoV, был обнаружен вирус-специфический Т-клеточный иммунитет (против белков S, N, M) спустя более 10 лет после перенесенного заболевания [1].
Поэтому более перспективной является стратегия использования в качестве Т-клеточных иммуногенов также последовательностей, входящих в состав минорных белков вируса, наименее подверженных изменчивости из-за меньшего давления отбора. Кроме того, перспективно рассматривать включение в состав Т-клеточного иммуногена убиквитин, способствующий более эффективной презентации антигена антигенпрезентирующими клетками.
Техническим результатом заявляемого изобретения является создание вакцинной плазмидной ДНК-конструкции pBSI-COV-Ub, которая обеспечивает более стойкий (длительный) индуцирующий SARS-CoV-2-специфический Т-клеточный иммунный ответ за счет включения высоко иммуногенных эпитопов из различных белков SARS-CoV-2 консервативных для разных штаммов этого вируса и рестриктируемых широким спектром аллелей MHC, способных индуцировать специфический цитотоксический и Т-хелперный иммунитет против различных штаммов вируса SARS-CoV-2.
Указанный технический результат достигается тем, что создан искусственный ген bsi-cov-ub, кодирующий искусственный Т-клеточный иммуноген BSI-COV-Ub, на N-конце которого расположена последовательность убиквитина, имеющий последовательность SEQ ID NO: 2 размером 1496 п.н. и содержащий на 5'-конце последовательность Козак CCGCCACC; между последовательностями, кодирующими убиквитин и BSI-COV расположен сайт для эндонуклеазы рестрикции PspLI, а на 3'-конце - два стоп-кодона TAATGA и сайт для эндонуклеазы рестрикции ApaI.
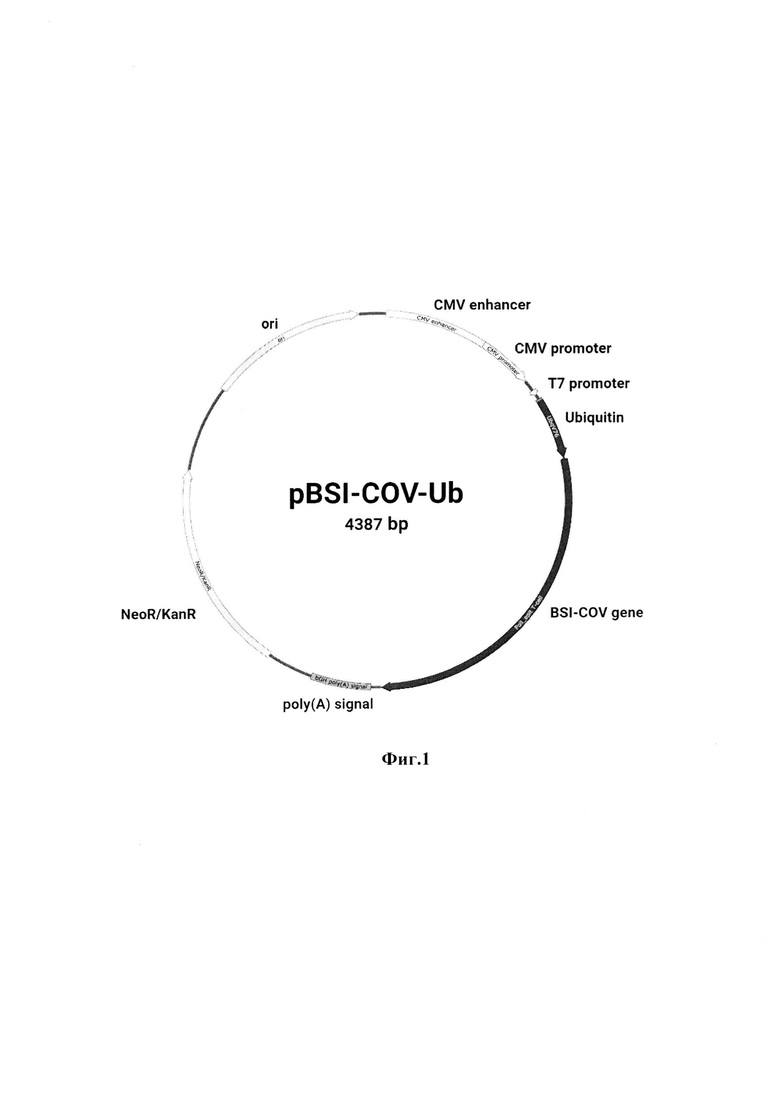
Указанный технический результат достигается также созданием плазмидной ДНК pBSI-COV-Ub, имеющей нуклеотидную последовательность SEQ ID NO: 1 размером 4387 п.н., молекулярный вес 2,7 МДа, содержащей целевой ген по п.1, кодирующий искусственный полиэпитопный белок-иммуноген BSI-COV-Ub с N-концевым убиквитином, перед которым находится промотор РНК-полимеразы фага Т7, обеспечивающий транскрипцию гена в эукариотической системе, и состоит из следующих фрагментов:- векторный фрагмент плазмиды pVAX, имеющий координаты 2180-709 и размер 2917 п.н., содержащий промотор РНК-полимеразы фага Т7, имеющий координаты 664-682 и размер 19 п.н., ген устойчивости к неомицину/канамицину NeoR/KanR, имеющий координаты 2614-3408 и размер 795 п.н., точку начала репликации ColE1 ori, имеющую координаты 3734-4322 и размер 589 п.н., а также энхансер и промотор цитомегаловируса, имеющие координаты 36-619 и размер 584 п.н.;
- фрагмент, содержащий ген убиквитина и имеющий координаты 710-938 и размер 229 п.н.;
- фрагмент, содержащий целевой искусственный ген bsi-cov и имеющий координаты 944-2179 и размер 1236 п.н.
Указанный технический результат достигается также созданием белка-иммуногена BSI-COV-Ub, имеющего последовательность SEQ ID NO: 3 длиной 490 а.к.о., содержащего N-концевой убиквитин, который увеличивает эффективность процессинга иммуногена, синтезированного в клетке, за счет нацеливания на протеасому.
При иммунизации животных созданной ДНК-конструкцией регистрируется специфический Т-клеточный ответ, более сильный по сравнению с конструкцией, кодирующей полиэпитопный иммуноген без убиквитина.
Изобретение иллюстрируется следующими графическими материалами.
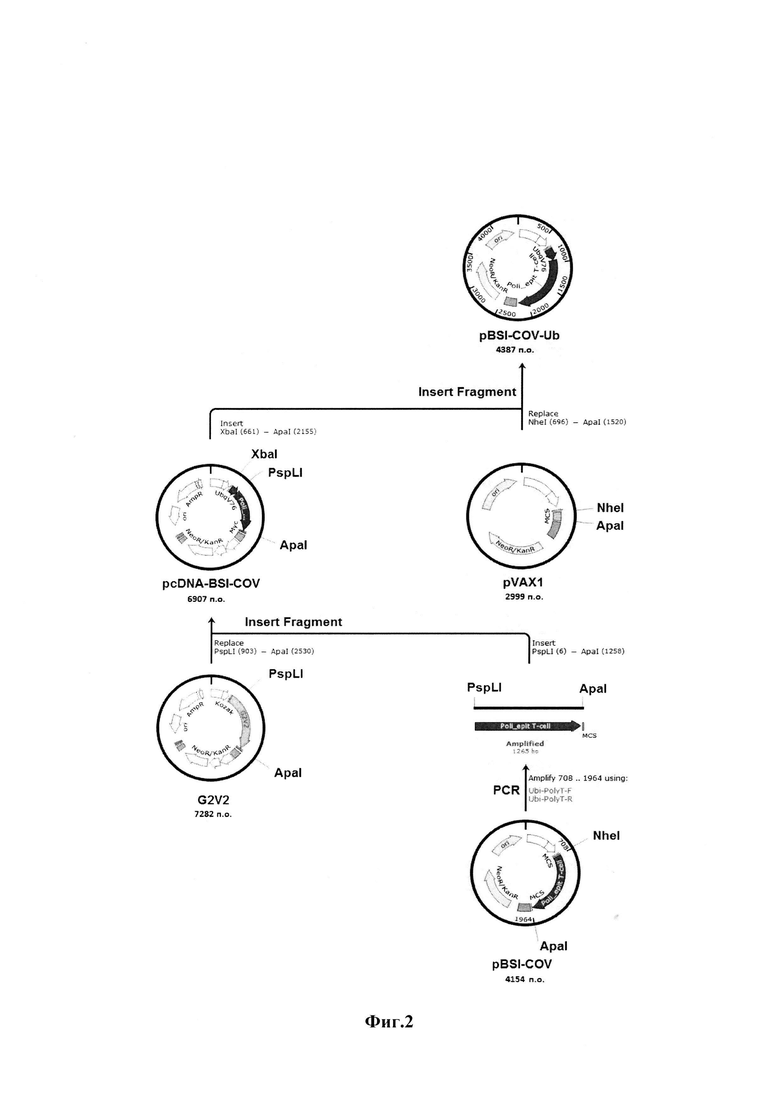
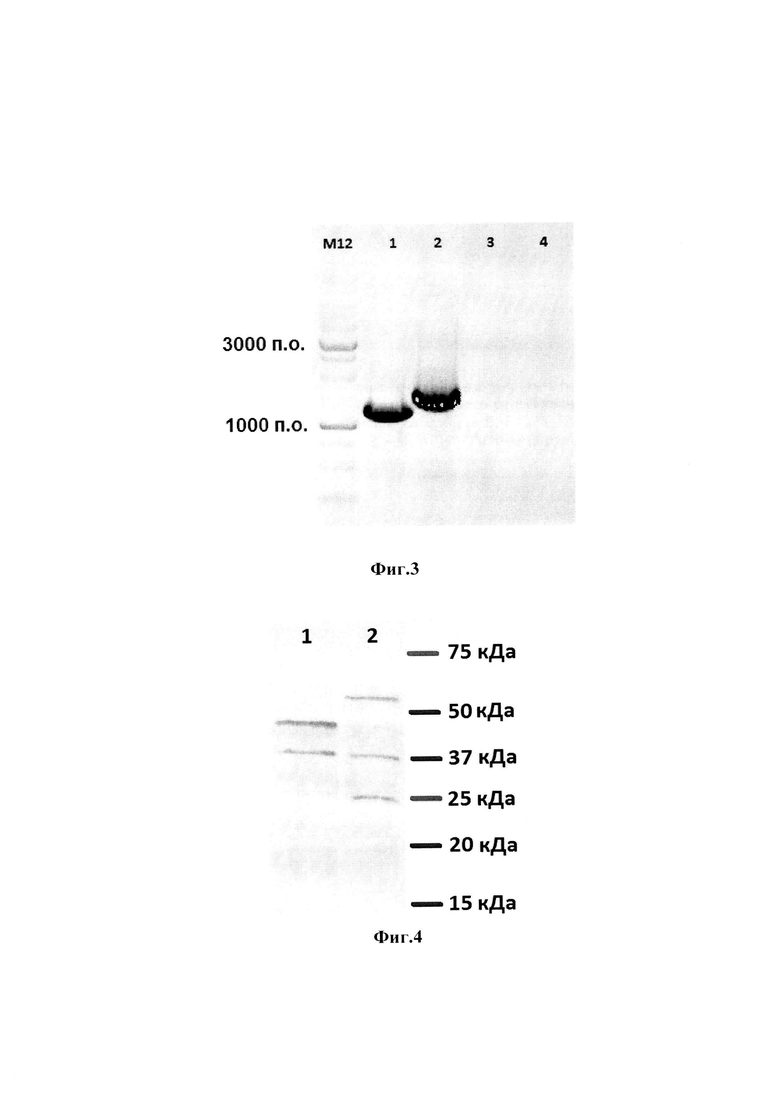
На фиг.1 приведена карта плазмиды pBSI-COV-Ub, а на фиг.2 - схема получения плазмиды pBSI-COV-Ub. На фиг.3 приведен электрофоретический анализ экспрессии гена bsi-cov в трансфицированных клетках HEK293T в 1% агарозном геле:
- дорожка М12 - маркеры молекулярной массы;
- дорожки 1 и 2 - продукты, полученные с помощью ОТ-ПЦР из тотальной РНК клеток HEK293T, трансфицированных pBSI-COV и pBSI-COV-Ub соответственно;
- дорожки 3 и 4 - продукты, полученный с помощью ПЦР из тотальной РНК клеток HEK293T, трансфицированных pBSI-COV и pBSI-COV-Ub соответственно, без стадии обратной транскрипции.
На фиг.4 представлен анализ продукции белков в клетках HEK293T с помощью вестерн-блоттинга и МКА 29F2 к маркерному эпитопу:
- дорожки 1 и 2 - лизаты HEK293T, трансфицированных pBSI-COV и pBSI-COV-Ub, соответственно;
- справа - маркер молекулярных масс Precision Plus Protein Dual Color Standards (Bio-rad, США).
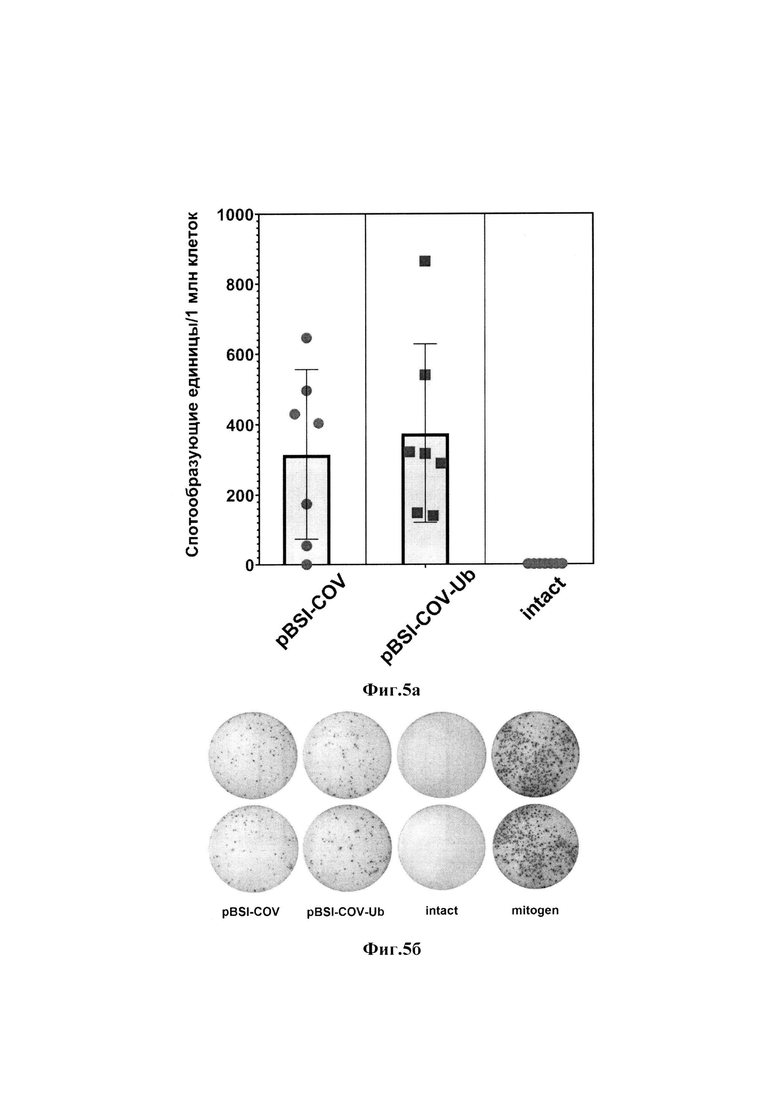
На фиг.5а приведены результаты оценки вирус-специфических Т-лимфоцитов у иммунизированных животных на 31 сутки после начала иммунизации с помощью метода ELISpot, а на фиг.5б - репрезентативные изображения лунок планшетов после проведения анализа ELISpot. На фиг.6а представлена доля SARS-CoV-2-специфических цитокин-продуцирующих CD4+и CD8+Т-клеток (по данным ICS и проточной цитометрии), а на фиг.6б - типичные диаграммы, полученные при анализе CD4+и CD8+Т-лимфоцитов с помощью ICS, проточного цитофлуориметра ZE5 и программного обеспечения Everest.
Ниже приведены примеры 1-5 описания заявляемых объектов изобретения.
Пример 1. Дизайн гена bsi-cov
Предсказание Т-клеточных эпитопов было выполнено с использованием программы NetMHCpan-4.1 и базы данных Immune Epitope Database 2.22, IEDB 2.22 (Табл. 1-3).
Перечисленные в табл.1 и 2 пептиды были проанализированы на консервативность для разных штаммов вируса SARS-CoV-2, в частности Ухань, Гамма, Дельта, Омикрон. Для каждого пептида была установлена более чем 87% гомологичность по отношению к белкам указанных штаммов SARS-CoV-2.
Предсказанные эпитопы объединили в кластеры, которые совпадают с фрагментами белков S, N, M и E (табл.4). Данные фрагменты, содержащие также большое количество эпитопов, рестриктируемых широким спектром аллелей HLA II, объединили в одну последовательность. К N-концу спроектированного белка был добавлен эпитоп PADRE, к C-концу - маркерный эпитоп EPFRDYVDRFYKTLR из белка p24 ВИЧ-1, узнаваемый моноклональными антителами 29F2.
В результате была спроектирована аминокислотная последовательность искусственного полиэпитопного Т-клеточного иммуногена, получившего название BSI-COV. Оптимизацию нуклеотидной последовательности гена, кодирующего белок BSI-COV, проводили с помощью программы Jcat (http://www.jcat.de) с учетом кодонов, обеспечивающих наиболее эффективную трансляцию в клетках млекопитающих.
Пример 2. Дизайн плазмиды pBSI-COV-Ub
Для получения конструкции, кодирующей полиэпитопный иммуноген с убиквитином на N-конце, была использована плазмида G2V2, ранее полученная в отделе биоинженерии, в которой закодирована генетическая конструкция иммуногена гриппа и убиквитин [14], а также плазмида pBSI-COV, кодирующая искусственный иммуноген SARS-CoV-2 без убиквитина. Схематически интересующий нас регион плазмиды G2V2 выглядит следующим образом: XbaI-Kozak-Ubq-PspLI-Gene-AgeI-Marker-Stop-ApaI. С помощью специально рассчитанных праймеров (Ubi-PolyT-F (прямой) - 5'-tttttCGTACGATGGCCAAGTTCGTGGCC-3', Ubi-PolyT-R (обратный) - 5'-TTAAACGGGCCCTCATTAGCGCA-3') и ПЦР в состав гена bsi-cov, был введен сайт рестрикции PspLI, в результате чего после рестрикции PspLI и ApaI и лигирования была получена конструкция, кодирующая BSI-COV с N-концевым убиквитином в составе pcDNA. Затем данная последовательность была клонирована в векторную плазмиду pVAX1 по сайтам XbaI/NheI и ApaI (фиг.2). Конструкция получила название pBSI-COV-Ub (фиг.1). Структуру полученной конструкции подтверждали секвенированием по методу Сэнгера в ЦКП “Геномика” (Россия).
Пример 3. Наработка и очистка рекомбинантных плазмид pBSI-COV и pBSI-COV-Ub
Клетки Escherichia coli Stbl3 трансформировали плазмидами pBSI-COV и pBSI-COV-Ub с помощью хлористого кальция. Наработку плазмидной ДНК для иммунизации проводили в 2.7 л питательной среды LB с добавлением канамицина (25 мкг/мл). Плазмидную ДНК выделяли и очищали с помощью набора EndoFree Plasmid Giga Kit (QIAGEN, Германия) согласно рекомендациям производителя. Данный набор позволяет очень эффективно очистить препарат плазмидной ДНК от бактериальных эндотоксинов. После очистки структуру плазмиды проверяли рестрикционным анализом, количество и чистоту - спектрофотометрией.
Пример 4. Проверка эффективности экспрессии целевых генов в условиях in vitro
4.1 Трансфекция клеток HEK293T
Перевиваемые клетки почечного эмбрионального эпителия человека HEK-293Т (Human embryonic kidney), получены из коллекции клеточных культур ГНЦ ВБ «Вектор». Культуру клеток HEK-293Т культивировали при температуре 37ºC, 80% относительной влажности воздуха и 5% содержании CO2. В качестве ростовой среды использовали среду DMEM с добавлением 10% термоинактивированной (30 мин при температуре 56°С) фетальной бычьей сыворотки (FBS), 2 мМ L-глутамина и антибиотика (гентамицин (50 мкг/мл)). Трансфекцию производили в 12-луночных планшетах с использованием реагента Matra-A, содержащего магнитные частицы, в соответствии с инструкцией производителя. По истечении 48 часов после трансформации клетки снимали с использованием раствора Версена и центрифугировали 5000 об/мин, осадок растворяли в 30 мкл PBS для дальнейшей работы.
4.2 Исследование экспрессии генов на уровне мРНК
Для проведения ОТ-ПЦР из трансфицированных клеток выделяли тотальную РНК с использованием набора RNeasy Mini Kit (QIAGEN, Германия) в соответствии с инструкцией производителя.
Анализ экспрессии гена был проведен с помощью обратной транскрипции с ПЦР в одной пробирке набором Биомастер ОТ-ПЦР-Экстра (Биолабмикс, Россия) с использованием специфических праймеров: Ubi-PolyT-F 5'-tttttCGTACGATGGCCAAGTTCGTGGCC-3(и Ubi-PolyT-R 5'- TTAAACGGGCCCTCATTAGCGCA-3((для pBSI-COV); pVAX-G2V2-F 5'- tttttgAATTCCCGCCACCATGcaaat-3(и Ubi-PolyT-R 5'- TTAAACGGGCCCTCATTAGCGCA-3((для pBSI-COV-Ub). Продукты ПЦР были проанализированы в 1% агарозном геле. На электрофореграмме (фиг.3) видно, что размеры амплифицированных фрагментов составляют примерно 1250 и 1500 п.о., что соответствует теоретически рассчитанным фрагментам при использовании указанных специфических праймеров.
4.3 Исследование продукции целевых белков
Лизаты трансфицированных клеток исследовали на наличие целевых белков с помощью иммуноблотинга с использованием МКА 29F2 к маркерному эпитопу. Полосы, соответствующие белкам с наибольшим весом, соответствуют теоретически рассчитанным продуктам генов bsi-cov и bsi-cov-ub (46 и 54,5 кДа соответственно). Наличие лесенки из дискретных белков свидетельствует об эффективном процессинге Т-клеточных иммуногенов в клетке (фиг.4).
Пример 5. Иммуногенность ДНК-вакцинных конструкций pBSI-COV и pBSI-COV-Ub
5.1 Иммунизация лабораторных животных, подготовка образцов для исследования
Работа с животными проводилась в соответствии с «Руководством по уходу и использованию лабораторных животных». Протоколы были одобрены Комитетом по уходу и использованию животных (IACUC) при Государственном научном центре вирусологии и биотехнологии «Вектор» (Протокол БЭК №1 от 21.03.2023).
Для оценки иммуногенности созданных конструкций использовали самок мышей BALB/c массой 16-18 г.Мышей разделили на три группы по 7 животных в каждой и иммунизировали следующим образом: группа pBSI-COV - 100 мкг плазмиды pBSI-COV в 50 мкл физиологического раствора; группа pBSI-COV-Ub - 100 мкг плазмиды pBSI-COV-Ub в 50 мкл физиологического раствора; группа intact - не иммунизированные мыши. Мышей иммунизировали дважды внутримышечно (в верхнюю часть бедра задней конечности) в дни 0 и 21 с помощью электропорации. Электропорацию проводили с использованием электропоратора CUY21 EDIT II и электродов-пинцетов LF650P5 диаметром 5 мм (BEX CO, LTD., Япония) и предваряющей общей анестезией 2,5% изофлураном. Использовали следующий протокол электропорации: постоянный ток прямоугольной формы прямой и обратной полярности с 3 импульсами с напряжением 12 вольт в течение 30 мс и интервалом 950 мл с ограничением по силе тока в 45 мА [15]. Спустя 10 дней после второй иммунизации у животных были взяты селезенки для исследования иммунного ответа.
Селезенки последовательно измельчали на нейлоновых фильтрах для клеток с диаметром пор 70 и 40 мкм (BD Falcon, США). После лизиса эритроцитов лизирующим буфером (Sigma, США) спленоциты дважды промывали полной средой RPMI и помещали в 1 мл среды RPMI с 2 мМ L-глутамина, 50 мкг/мл гентамицина и 10% FBS (Thermo Fisher Scientific, США). Клетки подсчитывали с использованием автоматического счетчика клеток TC20 (Bio-Rad, Hercules, CA, USA).
5.2 Исследование поствакцинального Т-клеточного ответа с помощью ELISpot
Анализ Т-клеточного иммунного ответа проводили с использованием набора Mouse IFN-(ELISpot HRP (Mabtech, Швеция) в соответствии с инструкциями производителя. Спленоциты пассировали в количестве 2,5*105 клеток/лунку и добавляли среду RPMI с 10% FBS (отрицательный контроль), или смесь пептидов (каждого в концентрации 20 мкг/мл), или конканавалин А (положительный контроль). Клетки инкубировали в течение 20 ч при 37°С в присутствии 5% СО2. Затем планшеты промывали и инкубировали с первичными антителами против IFN-γ. Планшеты снова промывали и затем инкубировали со вторичным антителом, конъюгированным со пероксидазой хрена. После промывки планшеты инкубировали с TMB. Количество клеток, продуцирующих IFN-γ, подсчитывали с использованием ELISpot ридера (Carl Zeiss, Оберкохен, Германия). Спленоциты, выделенные от иммунизированных мышей BALB/c, стимулировали пулом пептидов из последовательности S-белка SARS-CoV-2, рестриктированных главным комплексом гистосовместимости (MHC) класса I (H2-Dd, H-2-Kd и H- 2-Ld) и молекулы MHC класса II (H2-IAd и H2-IEd) мышей BALB/c (табл.5). Пептиды были подобраны в соответствии с эпитопами, входящими в состав иммуногена и предсказанными с использованием инструментов IEDB Analysis Resource и синтезированы AtaGenix Laboratories (Ухань, Китай); чистота пептидов>80%.
Данные IFN-γ-ELISpot показали, что через две недели после второй иммунизации наблюдалось формирование Т-клеточного иммунитета у мышей, которым вводили вакцинные конструкции. Так, в группе, иммунизированной pBSI-COV, среднее количество Т-лимфоцитов, секретирующих IFN-γ, в расчете на 106 спленоцитов составляло 315, а в группе, иммунизированной pBSI-COV-Ub, - 375 (фиг.5а,б). Значения в опытных группах достоверно отличались от контрольной группы.
5.3 Исследование поствакцинального Т-клеточного ответа с помощью ICS и проточной цитофлуориметрии
Для анализа CD4+и CD8+Т-лимфоцитов, реагирующих выбросом IFN-(при стимуляции вирус-специфическими пептидами, 5*105 клеток пассировали в лунки 96-луночных культуральных планшетов с круглым дном (Jet-BIOFIL, Китай) и стимулировали смесью пептидов, указанной в табл.5. Каждый пептид добавляли в концентрации 20 мкг/мл на лунку и клетки инкубировали в течение 4 ч при 37°С в присутствии 5% СО2 и еще 16 ч с брефельдином А (5 мкг/мл, GolgiPlug BD Biosciences). На следующий день клетки окрашивали анти-CD3, конъюгированным с Alexa Fluor 700 (Biolegend, США), анти-CD4, конъюгированным с BV785 (Biolegend, США), и анти-CD8, конъюгированным с FITC (Biolegend, США); фиксировали 1% параформальдегидом в PBS; и пермеабилизировали 0,5% Tween 20 в PBS. Затем клетки окрашивали для выявления внутриклеточных цитокинов анти-IFNγ-APC, анти-IL2-BV421, анти-TNFα-PE и анти-IL4-BV605 (Biolegend, США). Образцы анализировали с использованием проточного цитометра ZE5 (Bio-Rad) и программного обеспечения Everest.
Анализ Т-клеточного ответа с помощью метода ICS показал, что при иммунизации мышей ДНК-вакцинами pBSI-COV и pBSI-COV-Ub формируются как CD3+/CD8+, так и CD3+/CD4+лимфоциты, способные продуцировать цитокины после стимуляции вирусными пептидами (фиг.6а,б). Этот факт свидетельствует о формировании выраженного вирус-специфического Т-клеточного ответа. Значения в опытных группах достоверно отличались от контрольной группы. Сравнивая ответы на pBSI-COV и pBSI-COV-Ub можно заметить, что наиболее высокий процент IFN-γ-продуцирующих CD4+и CD8+Т-клеток наблюдается в группе, иммунизированной pBSI-COV-Ub, что коррелирует с данными ELISpot. Однако эти различия не достоверны.
Повышение продукции после стимуляции пептидами было показано и для цитокинов IL-2, IL-4 и TNF-α, что свидетельствует о формировании сбалансированного вирус-специфического Т-клеточного ответа, очень важного для противовирусной защиты.
Таким образом, преимущество заявляемой вакцинной платформы по сравнению с другими аналогами заключается в том, что, несмотря на смену антигенного профиля при появлении нового штамма и уменьшение нейтрализующей активности антител, Т-клеточный ответ, индуцированный искусственным белком-иммуногеном, составленным из консервативных эпитопов различных белков вируса, будет оставаться вирус-специфичным более длительное время. Поэтому вакцинная ДНК-конструкция pBSI-COV-Ub, кодирующая ген bsi-cov-ub, полезна в качестве компонента, который можно сочетать с любой другой вакциной, эффективно индуцирующей актуальный гуморальный ответ (субъединичные, инактивированные вакцины).
Список источников патентной и научно-технической информации
1. Ng O.W., Chia A., Tan A.T., Jadi R.S., Leong H.N., Bertoletti A., Tan Y.J. Memory T cell responses targeting the SARS coronavirus persist up to 11 years post-infection. Vaccine. 2016 Apr 12;34(17):2008-14. doi: 10.1016/j.vaccine.2016.02.063.
2. Walsh E.E., Frenck R.W. Jr, Falsey A.R., Kitchin N., Absalon J., Gurtman A., Lockhart S., Neuzil K., Mulligan M.J., Bailey R., Swanson K.A., Li P., Koury K., Kalina W., Cooper D., Fontes-Garfias C., Shi P.Y., Türeci Ö., Tompkins K.R., Lyke K.E., Raabe V., Dormitzer P.R., Jansen K.U., Şahin U., Gruber W.C. Safety and Immunogenicity of Two RNA-Based Covid-19 Vaccine Candidates. N Engl J Med. 2020 Dec 17;383(25):2439-2450. doi: 10.1056/NEJMoa2027906.
3. Baden L.R., El Sahly H.M., Essink B., Kotloff K., Frey S., Novak R., Diemert D., Spector S.A., Rouphael N., Creech C.B., McGettigan J., Khetan S., Segall N., Solis J., Brosz A., Fierro C., Schwartz H., Neuzil K., Corey L., Gilbert P., Janes H., Follmann D., Marovich M., Mascola J., Polakowski L., Ledgerwood J., Graham B.S., Bennett H., Pajon R., Knightly C., Leav B., Deng W., Zhou H., Han S., Ivarsson M., Miller J., Zaks T; COVE Study Group.Efficacy and Safety of the mRNA-1273 SARS-CoV-2 Vaccine. N Engl J Med. 2021 Feb 4;384(5):403-416. doi: 10.1056/NEJMoa2035389.
4. Stephenson K.E., Le Gars M., Sadoff J., de Groot A.M., Heerwegh D., Truyers C., Atyeo C., Loos C., Chandrashekar A., McMahan K., Tostanoski L.H., Yu J., Gebre M.S., Jacob-Dolan C., Li Z., Patel S., Peter L., Liu J., Borducchi E.N., Nkolola J.P., Souza M., Tan C.S., Zash R., Julg B., Nathavitharana R.R., Shapiro R.L., Azim A.A., Alonso C.D., Jaegle K., Ansel J.L., Kanjilal D.G., Guiney C.J., Bradshaw C., Tyler A., Makoni T., Yanosick K.E., Seaman M.S., Lauffenburger D.A., Alter G., Struyf F., Douoguih M., Van Hoof J., Schuitemaker H., Barouch D.H. Immunogenicity of the Ad26.COV2.S Vaccine for COVID-19. JAMA. 2021 Apr 20;325(15):1535-1544. doi: 10.1001/jama.2021.3645.
5. Logunov D.Y., Dolzhikova I.V., Shcheblyakov D.V., Tukhvatulin A.I., Zubkova O.V., Dzharullaeva A.S., Kovyrshina A.V., Lubenets N.L., Grousova D.M., Erokhova A.S., Botikov A.G., Izhaeva F.M., Popova O., Ozharovskaya T.A., Esmagambetov I.B., Favorskaya I.A., Zrelkin D.I., Voronina D.V., Shcherbinin D.N., Semikhin A.S., Simakova Y.V., Tokarskaya E.A., Egorova D.A., Shmarov M.M., Nikitenko N.A., Gushchin V.A., Smolyarchuk E.A., Zyryanov S.K., Borisevich S.V., Naroditsky B.S., Gintsburg A.L.; Gam-COVID-Vac Vaccine Trial Group.Safety and efficacy of an rAd26 and rAd5 vector-based heterologous prime-boost COVID-19 vaccine: an interim analysis of a randomised controlled phase 3 trial in Russia. Lancet. 2021 Feb 20;397(10275):671-681. doi: 10.1016/S0140-6736(21)00234-8.
6. Falsey A.R., Sobieszczyk M.E., Hirsch I., Sproule S., Robb M.L., Corey L., Neuzil K.M., Hahn W., Hunt J., Mulligan M.J., McEvoy C., DeJesus E., Hassman M., Little S.J., Pahud B.A., Durbin A., Pickrell P., Daar E.S., Bush L., Solis J., Carr Q.O., Oyedele T., Buchbinder S., Cowden J., Vargas S.L., Guerreros Benavides A., Call R., Keefer M.C., Kirkpatrick B.D., Pullman J., Tong T., Brewinski Isaacs M., Benkeser D., Janes H.E., Nason M.C., Green J.A., Kelly E.J., Maaske J., Mueller N., Shoemaker K., Takas T., Marshall R.P., Pangalos M.N., Villafana T., Gonzalez-Lopez A; AstraZeneca AZD1222 Clinical Study Group.Phase 3 Safety and Efficacy of AZD1222 (ChAdOx1 nCoV-19) Covid-19 Vaccine. N Engl J Med. 2021 Dec 16;385(25):2348-2360. doi: 10.1056/NEJMoa2105290.
7. Nathan A., Rossin E.J., Kaseke C., Park R.J., Khatri A., Koundakjian D., Urbach J.M., Singh N.K., Bashirova A., Tano-Menka R., Senjobe F., Waring M.T., Piechocka-Trocha A., Garcia-Beltran W.F., Iafrate A.J., Naranbhai V., Carrington M., Walker B.D., Gaiha G.D. Structure-guided T cell vaccine design for SARS-CoV-2 variants and sarbecoviruses. Cell. 2021 Aug 19;184(17):4401-4413.e10. doi: 10.1016/j.cell.2021.06.029.
8. Wang Q., Lei C., Wan H., Liu Q. Improved cellular immune response elicited by a ubiquitin-fused DNA vaccine against Mycobacterium tuberculosis. DNA Cell Biol. 2012 Apr;31(4):489-95. doi: 10.1089/dna.2011.1309.
9. Reguzova A., Antonets D., Karpenko L., Ilyichev A., Maksyutov R., Bazhan S. Design and evaluation of optimized artificial HIV-1 poly-T cell-epitope immunogens. PLoS One. 2015 Mar 18;10(3):e0116412. doi: 10.1371/journal.pone.0116412.
10. Zhuo M., Song L., Tang Y., Dai S., Chen X., Yu Y., Zang G., Tang Z. Vaccination with ubiquitin-hepatitis B core antigen-cytoplasmic transduction peptide enhances the hepatitis B virus-specific cytotoxic T-lymphocyte immune response and inhibits hepatitis B virus replication in transgenic mice. Mol Med Rep.2015 Sep;12(3):3591-3598. doi: 10.3892/mmr.2015.3834.
11. Dey A., Chozhavel Rajanathan T.M., Chandra H., Pericherla H.P.R., Kumar S., Choonia H.S., Bajpai M., Singh A.K., Sinha A., Saini G., Dalal P., Vandriwala S., Raheem M.A., Divate R.D., Navlani N.L., Sharma V., Parikh A., Prasath S., Sankar Rao M., Maithal K. Immunogenic potential of DNA vaccine candidate, ZyCoV-D against SARS-CoV-2 in animal models. Vaccine. 2021 Jul 5;39(30):4108-4116. doi: 10.1016/j.vaccine.2021.05.098.
12. Khobragade A., Bhate S., Ramaiah V., Deshpande S., Giri K., Phophle H., Supe P., Godara I., Revanna R., Nagarkar R., Sanmukhani J., Dey A., Rajanathan T.M.C., Kansagra K., Koradia P.; ZyCoV-D phase 3 Study Investigator Group.Efficacy, safety, and immunogenicity of the DNA SARS-CoV-2 vaccine (ZyCoV-D): the interim efficacy results of a phase 3, randomised, double-blind, placebo-controlled study in India. Lancet. 2022 Apr 2;399(10332):1313-1321. doi: 10.1016/S0140-6736(22)00151-9.
13. Safavi A., Kefayat A., Mahdevar E., Abiri A., Ghahremani F. Exploring the out of sight antigens of SARS-CoV-2 to design a candidate multi-epitope vaccine by utilizing immunoinformatics approaches. Vaccine. 2020 Nov 10;38(48):7612-7628. doi: 10.1016/j.vaccine.2020.10.016.
14. Bazhan S.I., Antonets D.V., Starostina E.V., Ilyicheva T.N., Kaplina O.N., Marchenko V.Y., Volkova O.Y., Bakulina A.Y., Karpenko L.I. In silico design of influenza a virus artificial epitope-based T-cell antigens and the evaluation of their immunogenicity in mice. J Biomol Struct Dyn. 2022 Apr;40(7):3196-3212. doi: 10.1080/07391102.2020.1845978.
15. Kisakov D.N., Kisakova L.A., Borgoyakova M.B., Starostina E.V., Taranov O.S., Ivleva E.K., Pyankov O.V., Zaykovskaya A.V., Shcherbakov D.N., Rudometov A.P., Rudometova N.B., Volkova N.V., Gureev V.N., Ilyichev A.A., Karpenko L.I. Optimization of In Vivo Electroporation Conditions and Delivery of DNA Vaccine Encoding SARS-CoV-2 RBD Using the Determined Protocol. Pharmaceutics. 2022 Oct 22;14(11):2259. doi: 10.3390/pharmaceutics14112259.
16. Международная заявка WO2021214703, МПК A61K 39/215, опубл. 28.10.2021 г.(прототип).
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="pBSI-CoV-Ub.xml"
softwareName="WIPO Sequence" softwareVersion="2.2.0"
productionDate="2023-04-19">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>1</ApplicationNumberText>
<FilingDate>2023-03-09</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>1</ApplicantFileReference>
<ApplicantName languageCode="ru">Федеральное бюджетное учреждение
науки «Государственный научный центр вирусологии и биотехнологии
«Вектор» Федеральной службы по надзору в сфере защиты прав
потребителей и благополучия человека (ФБУН ГНЦ ВБ «Вектор»
Роспотребнадзора)</ApplicantName>
<ApplicantNameLatin>State Research Center of Virology and
Biotechnology "Vector"</ApplicantNameLatin>
<InventionTitle languageCode="ru">Искусственный иммуноген
BSI-COV-Ub, содержащий убиквитин и эпитопы антигенов вируса
SARS-CoV-2 и индуцирующий SARS-CoV-2- специфический Т-клеточный
иммунитет, искусственный ген, кодирующий белок-иммуноген BSI-COV-Ub,
рекомбинантная плазмидная ДНК pBSI- COV-Ub, обеспечивающая экспрессию
целевого гена</InventionTitle>
<SequenceTotalQuantity>3</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>4387</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..4387</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>944..2179</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>gene of artificial multi-epitope T cell
immunogen</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>710..938</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>gene coding
ubiquitin</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>unsure</INSDFeature_key>
<INSDFeature_location>1..709</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>map</INSDQualifier_name>
<INSDQualifier_value>pVAX sequence</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>unsure</INSDFeature_key>
<INSDFeature_location>2180..4387</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>map</INSDQualifier_name>
<INSDQualifier_value>pVAX sequence</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gctgcttcgcgatgtacgggccagatatacgcgttgacattgattattg
actagttattaatagtaatcaattacggggtcattagttcatagcccatatatggagttccgcgttacat
aacttacggtaaatggcccgcctggctgaccgcccaacgacccccgcccattgacgtcaataatgacgta
tgttcccatagtaacgccaatagggactttccattgacgtcaatgggtggactatttacggtaaactgcc
cacttggcagtacatcaagtgtatcatatgccaagtacgccccctattgacgtcaatgacggtaaatggc
ccgcctggcattatgcccagtacatgaccttatgggactttcctacttggcagtacatctacgtattagt
catcgctattaccatggtgatgcggttttggcagtacatcaatgggcgtggatagcggtttgactcacgg
ggatttccaagtctccaccccattgacgtcaatgggagtttgttttggcaccaaaatcaacgggactttc
caaaatgtcgtaacaactccgccccattgacgcaaatgggcggtaggcgtgtacggtgggaggtctatat
aagcagagctctctggctaactagagaacccactgcttactggcttatcgaaattaatacgactcactat
agggagacccaagctggctagaccgccaccatgcaaatctttgtaaaaaccctgaccggaaagaccatca
ccttggaagtggaacctagtgacactattgaaaatgttaaggctaagatacaggataaagaggggatccc
tcctgaccagcaacggctcatctttgctggtaaacagctcgaggacggacggactctgtctgattacaat
atccagaaggaaagcaccctacacctggttctaagactgcgaggtgtacgtacgatggccaagttcgtgg
ccgcctggaccctgaaggccgccgccaagaagaacatcgccgactacaactacaagctgcccgacgactt
caccggctgcgtgatcgcctggaacagcaacaacctggacagcaaggtgggcggcaactacaactacctg
taccgcctgttccgcaagagcaacctgaagcccttcgagagcatcatcgcctacaccatgagcctgggcg
ccgagaacagcgtggcctacagcaacaacagcatcgccatccccaccaacttcaccatcggcctgaccgt
gctgccccccctgctgaccgacgagatgatcgcccagtacaccagcgccctgctggccggcaccatcacc
agcggctggaccttcggcgccggcgccgcccacctgatgagcttcccccagagcgccccccacggcgtgg
tgttcctgcacgtgacctacgtgcccgcccaggagaagaacctgaacgagagcctgatcgacctgcagga
gctgggcaagtacgagcagtacatcaagtggccctggtacatctggctgggcttcatcgccggcctgatc
gccatcgtgatggtgaccatcatgagcctggtgaagcccagcttctacgtgtacagccgcgtgaagaacc
tgaacagcagccgcgtgcccgacctgctggtgaccctggcctgcttcgtgctggccgccgtgtaccgcat
caactggatcaccggcggcatcgccatcgccatggcctgcctggtgggcctgaaggagatcaccgtggcc
accagccgcaccctgagctactacaagctgggcgccagccagcgcgtggccggcgacagcggcttcgccg
cctacttcccccgcggccagggcgtgcccatcaacaccaacagcagccccgacgaccagatcggctacta
ccgccgcgccacccgccgcatccgcggcggcgacggcaagtggccccagatcgcccagttcgcccccagc
gccagcgccttcttcggcatgagccgcatcggcatggaggtgacccccagcggcacctggctgacctaca
ccggcgccatcaagctggacgacaaggaccccaacttcaaggaccaggtgatcctgctgaacaagcacat
cgacgcctacaagaccttcccccccaccgagcccaagaaggacaagaagaagaaggagcccttccgcgac
tacgtggaccgcttctacaagaccctgcgctaatgagggcccgtttaaacccgctgatcagcctcgactg
tgccttctagttgccagccatctgttgtttgcccctcccccgtgccttccttgaccctggaaggtgccac
tcccactgtcctttcctaataaaatgaggaaattgcatcgcattgtctgagtaggtgtcattctattctg
gggggtggggtggggcaggacagcaagggggaggattgggaagacaatagcaggcatgctggggatgcgg
tgggctctatggcttctactgggcggttttatggacagcaagcgaaccggaattgccagctggggcgccc
tctggtaaggttgggaagccctgcaaagtaaactggatggctttctcgccgccaaggatctgatggcgca
ggggatcaagctctgatcaagagacaggatgaggatcgtttcgcatgattgaacaagatggattgcacgc
aggttctccggccgcttgggtggagaggctattcggctatgactgggcacaacagacaatcggctgctct
gatgccgccgtgttccggctgtcagcgcaggggcgcccggttctttttgtcaagaccgacctgtccggtg
ccctgaatgaactgcaagacgaggcagcgcggctatcgtggctggccacgacgggcgttccttgcgcagc
tgtgctcgacgttgtcactgaagcgggaagggactggctgctattgggcgaagtgccggggcaggatctc
ctgtcatctcaccttgctcctgccgagaaagtatccatcatggctgatgcaatgcggcggctgcatacgc
ttgatccggctacctgcccattcgaccaccaagcgaaacatcgcatcgagcgagcacgtactcggatgga
agccggtcttgtcgatcaggatgatctggacgaagagcatcaggggctcgcgccagccgaactgttcgcc
aggctcaaggcgagcatgcccgacggcgaggatctcgtcgtgacccatggcgatgcctgcttgccgaata
tcatggtggaaaatggccgcttttctggattcatcgactgtggccggctgggtgtggcggaccgctatca
ggacatagcgttggctacccgtgatattgctgaagagcttggcggcgaatgggctgaccgcttcctcgtg
ctttacggtatcgccgctcccgattcgcagcgcatcgccttctatcgccttcttgacgagttcttctgaa
ttattaacgcttacaatttcctgatgcggtattttctccttacgcatctgtgcggtatttcacaccgcat
acaggtggcacttttcggggaaatgtgcgcggaacccctatttgtttatttttctaaatacattcaaata
tgtatccgctcatgagacaataaccctgataaatgcttcaataatagcacgtgctaaaacttcattttta
atttaaaaggatctaggtgaagatcctttttgataatctcatgaccaaaatcccttaacgtgagttttcg
ttccactgagcgtcagaccccgtagaaaagatcaaaggatcttcttgagatcctttttttctgcgcgtaa
tctgctgcttgcaaacaaaaaaaccaccgctaccagcggtggtttgtttgccggatcaagagctaccaac
tctttttccgaaggtaactggcttcagcagagcgcagataccaaatactgtccttctagtgtagccgtag
ttaggccaccacttcaagaactctgtagcaccgcctacatacctcgctctgctaatcctgttaccagtgg
ctgctgccagtggcgataagtcgtgtcttaccgggttggactcaagacgatagttaccggataaggcgca
gcggtcgggctgaacggggggttcgtgcacacagcccagcttggagcgaacgacctacaccgaactgaga
tacctacagcgtgagctatgagaaagcgccacgcttcccgaagggagaaaggcggacaggtatccggtaa
gcggcagggtcggaacaggagagcgcacgagggagcttccagggggaaacgcctggtatctttatagtcc
tgtcgggtttcgccacctctgacttgagcgtcgatttttgtgatgctcgtcaggggggcggagcctatgg
aaaaacgccagcaacgcggcctttttacggttcctggccttttgctggccttttgctcacatgttctt</
INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>1496</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..1496</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>249..1484</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>bsi-cov</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>15..243</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>ubiquitin</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gctagaccgccaccatgcaaatctttgtaaaaaccctgaccggaaagac
catcaccttggaagtggaacctagtgacactattgaaaatgttaaggctaagatacaggataaagagggg
atccctcctgaccagcaacggctcatctttgctggtaaacagctcgaggacggacggactctgtctgatt
acaatatccagaaggaaagcaccctacacctggttctaagactgcgaggtgtacgtacgatggccaagtt
cgtggccgcctggaccctgaaggccgccgccaagaagaacatcgccgactacaactacaagctgcccgac
gacttcaccggctgcgtgatcgcctggaacagcaacaacctggacagcaaggtgggcggcaactacaact
acctgtaccgcctgttccgcaagagcaacctgaagcccttcgagagcatcatcgcctacaccatgagcct
gggcgccgagaacagcgtggcctacagcaacaacagcatcgccatccccaccaacttcaccatcggcctg
accgtgctgccccccctgctgaccgacgagatgatcgcccagtacaccagcgccctgctggccggcacca
tcaccagcggctggaccttcggcgccggcgccgcccacctgatgagcttcccccagagcgccccccacgg
cgtggtgttcctgcacgtgacctacgtgcccgcccaggagaagaacctgaacgagagcctgatcgacctg
caggagctgggcaagtacgagcagtacatcaagtggccctggtacatctggctgggcttcatcgccggcc
tgatcgccatcgtgatggtgaccatcatgagcctggtgaagcccagcttctacgtgtacagccgcgtgaa
gaacctgaacagcagccgcgtgcccgacctgctggtgaccctggcctgcttcgtgctggccgccgtgtac
cgcatcaactggatcaccggcggcatcgccatcgccatggcctgcctggtgggcctgaaggagatcaccg
tggccaccagccgcaccctgagctactacaagctgggcgccagccagcgcgtggccggcgacagcggctt
cgccgcctacttcccccgcggccagggcgtgcccatcaacaccaacagcagccccgacgaccagatcggc
tactaccgccgcgccacccgccgcatccgcggcggcgacggcaagtggccccagatcgcccagttcgccc
ccagcgccagcgccttcttcggcatgagccgcatcggcatggaggtgacccccagcggcacctggctgac
ctacaccggcgccatcaagctggacgacaaggaccccaacttcaaggaccaggtgatcctgctgaacaag
cacatcgacgcctacaagaccttcccccccaccgagcccaagaaggacaagaagaagaaggagcccttcc
gcgactacgtggaccgcttctacaagaccctgcgctaatgagggccc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>490</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..490</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q3">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MQIFVKTLTGKTITLEVEPSDTIENVKAKIQDKEGIPPDQQRLIFAGKQ
LEDGRTLSDYNIQKESTLHLVLRLRGVRTMAKFVAAWTLKAAAKKNIADYNYKLPDDFTGCVIAWNSNNL
DSKVGGNYNYLYRLFRKSNLKPFESIIAYTMSLGAENSVAYSNNSIAIPTNFTIGLTVLPPLLTDEMIAQ
YTSALLAGTITSGWTFGAGAAHLMSFPQSAPHGVVFLHVTYVPAQEKNLNESLIDLQELGKYEQYIKWPW
YIWLGFIAGLIAIVMVTIMSLVKPSFYVYSRVKNLNSSRVPDLLVTLACFVLAAVYRINWITGGIAIAMA
CLVGLKEITVATSRTLSYYKLGASQRVAGDSGFAAYFPRGQGVPINTNSSPDDQIGYYRRATRRIRGGDG
KWPQIAQFAPSASAFFGMSRIGMEVTPSGTWLTYTGAIKLDDKDPNFKDQVILLNKHIDAYKTFPPTEPK
KDKKKKEPFRDYVDRFYKTLR</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Искусственный ген, кодирующий белок-иммуноген BSI-COV, рекомбинантная плазмидная ДНК pBSI-COV, обеспечивающая экспрессию целевого гена, и искусственный полиэпитопный белок-иммуноген BSI-COV, содержащий эпитопы антигенов вируса SARS-CoV-2 и индуцирующий SARS-CoV-2-специфический T-клеточный иммунитет | 2023 |
|
RU2806556C1 |
| Плазмидная ДНК-матрица и молекула мРНК, используемая для создания вакцины против коронавируса, полученная с использованием ДНК-матрицы и обеспечивающая экспрессию гена искусственного полиэпитопного белка-иммуногена, содержащего консервативные эпитопы антигенов вируса SARS-Cov-2 и индуцирующего SARS-Cov-2-специфический иммунитет | 2024 |
|
RU2839372C1 |
| ИСКУССТВЕННЫЙ БЕЛОК-ИММУНОГЕН TCI, СОДЕРЖАЩИЙ МНОЖЕСТВЕННЫЕ CTL-ЭПИТОПЫ ОСНОВНЫХ АНТИГЕНОВ ВИЧ-1, ИСКУССТВЕННЫЙ ГЕН TCI, КОДИРУЮЩИЙ ПОЛИЭПИТОПНЫЙ БЕЛОК-ИММУНОГЕН TCI | 2002 |
|
RU2238946C2 |
| Искусственный ген MEL-TCI, кодирующий полиэпитопный белок-иммуноген MEL-TCI, рекомбинантная плазмидная ДНК pMEL-TCI, обеспечивающая экспрессию искусственного гена MEL-TCI и искусственный белок-иммуноген MEL-TCI, содержащий CTL- и Th-эпитопы антигенов меланомы, рестриктированные множественными аллелями HLA I и II класса | 2017 |
|
RU2650872C1 |
| ИСКУССТВЕННЫЙ ГЕН MEL-TCI-A0201, КОДИРУЮЩИЙ ПОЛИЭПИТОПНЫЙ БЕЛОК-ИММУНОГЕН MEL-TCI-A0201, РЕКОМБИНАНТНАЯ ПЛАЗМИДНАЯ ДНК pMEL-TCI-A0201, ОБЕСПЕЧИВАЮЩАЯ ЭКСПРЕССИЮ ИСКУССТВЕННОГО ГЕНА MEL-TCI-A0201 И ИСКУССТВЕННЫЙ БЕЛОК-ИММУНОГЕН MEL-TCI-A0201, СОДЕРЖАЩИЙ МНОЖЕСТВЕННЫЕ CTL- И Th-ЭПИТОПЫ АНТИГЕНОВ МЕЛАНОМЫ | 2012 |
|
RU2522830C2 |
| Искусственный ген N1new, кодирующий нуклеокапсидный белок коронавируса SARS-CoV-2, и рекомбинантная плазмида pET-28a-N1new, обеспечивающая экспрессию искусственного гена | 2021 |
|
RU2762962C1 |
| Искусственные гены, кодирующие белки-иммуногены EV.CTL и EV.Th, рекомбинантные плазмидные ДНК pEV.CTL и pEV.Th, обеспечивающие экспрессию искусственных генов, и искусственные Т-клеточные полиэпитопные белки-иммуногены EV.CTL и EV.Th, содержащие эпитопы антигенов вируса Эбола, используемые для создания вакцины против вируса Эбола | 2018 |
|
RU2713723C1 |
| Конъюгат белка рецепторсвязывающего домена (RBD) поверхностного гликопротеина S вируса SARS-CoV-2 с полимером полиглюкин-спермидин (PGS) и вакцинный комплекс против коронавирусной инфекции COVID-19 на основе указанного конъюгата и плазмидной ДНК pVAX-RBD | 2022 |
|
RU2781294C1 |
| Интегративный плазмидный вектор pVEAL3-RBDdel, обеспечивающий синтез и секрецию рекомбинантного белка рецепторсвязывающего домена RBDdelta коронавируса SARS-CoV-2 в клетках млекопитающих, рекомбинантный штамм клеточной линии CHO-K1- RBDdelta и рекомбинантный белок RBDdelta SARS-CoV-2, продуцируемый штаммом клеточной линии | 2023 |
|
RU2816175C1 |
| Рекомбинантный штамм Sen-Sdelta(M) вируса Сендай, продуцирующий S белок SARS-CoV-2 и используемый для получения интраназальной вакцины против COVID-19 | 2024 |
|
RU2837536C1 |

Изобретение относится к биотехнологии. Описан искусственный ген bsi-cov-ub, кодирующий искусственный полиэпитопный Т-клеточный белок-иммуноген BSI-COV-Ub, имеющий нуклеотидную последовательность SEQ ID NO: 2, содержащий на 5'-конце последовательность Козак CCGCCACC; между последовательностями, кодирующими убиквитин и BSI-COV расположен сайт для эндонуклеазы рестрикции PspLI, а на 3'-конце - два стоп-кодона TAATGA и сайт для эндонуклеазы рестрикции ApaI. Также описана рекомбинантная плазмидная ДНК pBSI-COV-Ub, имеющая нуклеотидную последовательность SEQ ID NO: 1. Кроме того, описан полиэпитопный белок-иммуноген BSI-COV-Ub, имеющий аминокислотную последовательность SEQ ID NO: 3, содержащий консервативные эпитопы из S, E, M, N белков вируса SARS-CoV-2, универсальный Т-хелперный эпитоп PADRE, маркерный эпитоп, связывающийся с известными МКА 29F2, и N-концевой убиквитин, увеличивающий эффективность процессинга иммуногена, синтезированного в клетке, за счет нацеливания на протеасому и индуцирующий SARS-CoV-2-специфический Т-клеточный иммунитет. Техническим результатом является создание безопасной и эффективной полиэпитопной Т-клеточной вакцинной ДНК-конструкции, способной индуцировать специфический цитотоксический и Т-хелперный ответ против различных штаммов вируса SARS-CoV-2. 3 н.п. ф-лы, 8 ил., 5 табл., 5 пр.
1. Искусственный ген bsi-cov-ub, кодирующий искусственный полиэпитопный Т-клеточный белок-иммуноген BSI-COV-Ub, имеющий нуклеотидную последовательность SEQ ID NO: 2 размером 1489 п. н. и содержащий на 5'-конце последовательность Козак CCGCCACC; между последовательностями, кодирующими убиквитин и BSI-COV расположен сайт для эндонуклеазы рестрикции PspLI, а на 3'-конце - два стоп-кодона TAATGA и сайт для эндонуклеазы рестрикции ApaI.
2. Рекомбинантная плазмидная ДНК pBSI-COV-Ub, имеющая размер 4387 п. н., молекулярный вес 2,7 МДа и нуклеотидную последовательность SEQ ID NO: 1, содержащая целевой ген по п. 1, кодирующий искусственный полиэпитопный белок BSI-COV-Ub, содержащий на N-конце убиквитин, перед которым находится промотор РНК-полимеразы фага Т7, обеспечивающий транскрипцию гена в эукариотической системе, и состоящая из следующих фрагментов:
- векторный фрагмент плазмиды pVAX размером 2917 п. н., имеющий координаты 2179-710, содержащий промотор РНК-полимеразы фага Т7 с координатами 664-682 и размером 19 п. н., ген устойчивости к неомицину/канамицину NeoR/KanR с координатами 2614-3408 и размером 795 п. н., точку начала репликации ColE1 ori с координатами 3734-4322 и размером 589 п. н., а также энхансер и промотор цитомегаловируса, имеющий координаты 36-619 и размер 584 п. н.;
- фрагмент, имеющий координаты 710-938 и размер 229 п. н. и содержащий ген убиквитина;
- фрагмент, имеющий координаты 944-2179 и размер 1236 п. н. и содержащий целевой искусственный ген BSI-COV по п. 1.
3. Полиэпитопный белок-иммуноген BSI-COV-Ub, имеющий аминокислотную последовательность SEQ ID NO: 3 длиной 490 а.к.о., содержащий консервативные эпитопы из S, E, M, N белков вируса SARS-CoV-2, универсальный Т-хелперный эпитоп PADRE, маркерный эпитоп, связывающийся с известными МКА 29F2 и N-концевой убиквитин, увеличивающий эффективность процессинга иммуногена, синтезированного в клетке, за счет нацеливания на протеасому и индуцирующий SARS-CoV-2-специфический Т-клеточный иммунитет.
| WO 2021214703 A1, 28.10.2021 | |||
| Рекомбинантные вирусоподобные частицы для индукции специфического иммунитета против вируса тяжелого острого респираторного синдрома SARS-CoV-2 | 2021 |
|
RU2769224C1 |
| WO 2021175960 A1, 10.09.2021. | |||
Авторы
Даты
2023-11-01—Публикация
2023-05-29—Подача