Изобретение относится к полинуклеотидному экспрессионному вектору, содержащему 5'НТО, 3'НТО и поли(А)хвост, и может быть использовано для получения матриц для in vitro синтеза мРНК, обладающих увеличенной трансляционной эффективностью в эукариотических клетках и может быть использована в генетической инженерии, биотехнологии и медицине для производства вакцинных препаратов против SARS-CoV-2.
Распространение высокопатогенных и пандемических опасных вирусов стимулировало разработку мРНК-вакцин. Поскольку такая платформа позволяет очень быстро перейти на выпуск массового производства новой вакцины с учетом актуальных штаммов вируса. Важным этапом при конструировании мРНК-вакцин является оптимизация ее нуклеотидной последовательности как кодирующей, так и некодирующих частей, которые могут оказывать влияние на скорость инициации трансляции и деградации мРНК. В значительной степени эффективность трансляции мРНК и ее устойчивость к деградации зависит от нетранслируемых областей (НТО), которые представляют собой некодирующие части последовательности мРНК, расположенные выше (5'НТО) и ниже (3'НТО) кодирующей области мРНК. Нуклеотидные и структурные особенности 5'-НТО обусловливают связывание с ней различных белковых факторов, активирующих или подавляющих трансляцию. При проектировании мРНК-вакцин 5'-НТО рассматривают как регуляторный элемент, который потенциально может обеспечить высокоэффективную экспрессию и синтез целевого белка в эукариотической клетке. Однако, на данный момент сложно предсказать регуляторный потенциал какой-либо 5'НТО, исходя только из ее последовательности. Актуальным является поиск оптимальных комбинаций 5'НТО и 3'НТО, которые являются более эффективными. Все это может повысить иммуногенность мРНК-вакцин, помочь снизить их дозу и уменьшить побочные эффекты.
За последние несколько лет в области вакцин на основе нуклеиновых кислот было достигнуты впечатляющие успехи. мРНК-вакцины становятся перспективной альтернативой другим типам вакцин, основанных на живом или инактивированном вирусе, векторных вакцин, субъединичных белках, вирусоподобных частицах и др. Эти вакцины продемонстрировали высокую эффективность (способность индуцировать как гуморальный, так и клеточный иммунитет) и весьма приемлемую безопасность. Конструирование плазмиды, получение штамма-продуцента, ферментацию и очистку плазмиды можно осуществить в течение 2-4 недель. Технология производства таких вакцин не требует сложных манипуляций или работы с опасными патогенами, что значительно облегчает процесс их создания и снижает его общую стоимость.
Известна разработка научно-технологического университета "Сириус" полинуклеотидного компонента 5'UTR эукариотического экспрессионного вектора для повышения экспрессии представляющей интерес последовательности, который содержит лидерную регуляторную последовательность TPL (состоящую из трех частей лидерную последовательность) с SEQ ID NO: 5, а также плазмидный эукариотический экспрессионный вектор, кодирующий полинуклеотидный компонент 5'UTR по п. 1. (патент РФ №2804421, МПК C12N 15/11, опубл. 29.09.2023 г.). Патент содержит также применение плазмидного экспрессионного вектора из п. 2 для in vitro синтеза РНК.
Наиболее близким аналогом (прототипом) является вектор на основе мРНК для увеличенной продукции целевого белка, содержащий 5'-нетраслируемую область, выбранную из SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, SEQ ID NO: 4, область, кодирующую целевой белок, и 3'-нетраслируемую область, выбранную из SEQ ID NO: 5, или
вектор на основе мРНК для увеличенной продукции целевого белка, содержащий 5'-нетраслируемую область, выбранную из SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, область, кодирующую целевой белок, и 3'-нетраслируемую область, выбранную из SEQ ID NO: 6 (патент РФ №2792231, МПК C12N 15/67, опубл. 21.03.2023 г.). Область, кодирующая целевой белок, представляет собой последовательность, кодирующую вакцинный антиген или область, кодирующая целевой белок, представляет собой последовательность, кодирующую антитело, или область, кодирующая целевой белок, представляет собой последовательность, кодирующую терапевтический белок, или область, кодирующая целевой белок, представляет собой последовательность, кодирующую фермент. Вектор используют для синтеза целевого белка в клетках млекопитающих. Синтез целевого белка происходит in vivo.
В приведенных выше известных конструкциях обеспечивается повышение уровня экспрессии мРНК.
В заявляемом техническом решении осуществлена концепция быстрого производства мРНК-вакцины за счет создания платформы в виде ДНК-кассеты, несущей в себе более эффективные регуляторные нетранслируемые области (НТО) последовательности, и другие необходимые для функционирования мРНК элементы, такие как кэп и поли(А)хвост. Данная конструкция позволит в кратчайшие сроки встроить последовательность целевого белка и получить ДНК-матрицу для синтеза желаемой мРНК.
Таким образом, поиск наиболее эффективных НТО повысит эффективность мРНК-вакцин, а использование ДНК-кассеты, позволяющей встраивать любые целевые гены, позволит быстро получить матрицу для синтеза мРНК.
Техническим результатом заявляемого изобретения является создание платформы в виде ДНК-кассеты и ДНК-матрицы на ее основе для синтеза мРНК, содержащие все необходимые элементы, важные для эффективной работы мРНК-вакцины, в том числе сайты эндонуклеаз рестрикции, позволяющие встроить в данную конструкцию последовательность любого белка-антигена, который будет синтезировать с высоким уровнем экспрессии мРНК-вакцина в эукариотических клетках.
Указанный технический результат достигается тем, что создана плазмидная ДНК-кассета pVAX-C3-PolyA, имеющая нуклеотидную последовательность SEQ ID NO: 1 размером 3171 п.н., молекулярный вес 1,9 МДа и содержащая в соответствии с физической и генетической картой на фиг. 8 элементы, необходимые для синтеза мРНК: модифицированный Т7 промотор с инициирующими нуклеотидами AGG, 5'НТО ChM, 3'НТО β-глобина, поли(А)хвост длиной 100 нуклеотидов с внутренним линкером:
- векторный фрагмент ДНК-плазмиды pVAX, имеющий размер 2837 п.н. и координаты 958-624;
- ген устойчивости к неомицину/канамицину (NeoR/KanR) с координатами 1398-2192 размером 795 п.н. и точка начала репликации ColEl ori с координатами 2518-3106 размером 589 п.н., обеспечивающие селекцию и амплификацию целевой плазмиды в клетках бактерий Escherichia coli;
- фрагмент размером 20 п.н., имеющий координаты 625-644, являющийся промотором РНК-полимеразы фага Т7, с инициирующими нуклеотидами AGG и необходимый для использования;
- фрагмент размером 56 п.н., имеющий координаты 645-700, являющийся модифицированной химерной последовательностью (ChM) и используемый в качестве 5'НТО для мРНК-вакцины против COVID-19;
- фрагмент размером 135 п.н., имеющий координаты 707-841, являющийся последовательностью β-глобина человека и используемый в качестве 3'НТО;
- фрагмент размером 111 п.н., имеющий координаты 848-958, представляющий собой поли(А)хвост длиной 100 нуклеотидов с внутренним линкером из 10 нуклеотидов 30(A)GCATATGACT70(A);
- сайты эндонуклеазы рестрикции CciNI и BamHI, имеющие координаты 690 и 701 соответственно;
- сайт эндонуклеазы рестрикции Bso31I, имеющий координаты 958.
Указанный технический результат достигается также тем, что создана плазмидная ДНК-матрица pVAX-C3-RBD-PolyA для синтеза мРНК, полученная с использованием плазмидной ДНК-кассеты pVAX-C3-PolyA по п. 1, имеющая нуклеотидную последовательность SEQ ID NO: 5 размером 3903 п.н., молекулярный вес 2,3 МДа, включающая целевой ген, кодирующий химерный белок 176-RBD, имеющий аминокислотную последовательность SEQ ID NO: 7 и координаты 701 по 1432 и состоящей в соответствии с физической и генетической картой на фиг. 9 из следующих фрагментов:
- векторный фрагмент ДНК-плазмиды pVAX, имеющий размер 2837 п.н. и координаты 1690-624;
- ген устойчивости к неомицину/канамицину (NeoR/KanR), имеющий координаты 2130-2924 и размер 795 п.н., и точка начала репликации ColEl ori, имеющая координаты 3250-3838 и размер 589 п.н., обеспечивающие селекцию и амплификацию целевой плазмиды в клетках бактерий Escherichia coli;
- фрагмент размером 20 п.н., имеющий координаты 625-644 и являющийся промотором РНК-полимеразы фага Т7, с инициирующими нуклеотидами AGG, обеспечивающий транскрипцию гена, кодирующего химерный белок 176-RBD;
- фрагмент размером 56 п.н., имеющий координаты 645-700, являющийся модифицированной химерной последовательностью (ChM) и используемый в качестве 5'НТО для мРНК-вакцины против COVID-19;
- фрагмент размером 732 п.н., имеющий координаты 701-1432, содержащий искусственный ген 176-RBD с инициирующим кодоном ATG;
- фрагмент размером 135 п.н., имеющий координаты 1439-1573, являющийся последовательностью β-глобина человека и используемый в качестве 3 'НТО;
- фрагмент размером 111 п.н., имеющий координаты 1580-1690, представляющий собой поли(А)хвост длиной 100 нуклеотидов с внутренним линкером из 10 нуклеотидов 30(A)GCATATGACT70(A);
- сайты эндонуклеазы рестрикции CciNI и BamHI, имеющие координаты 690 и 1433 соответственно;
- сайт эндонуклеазы рестрикции Bso31I, имеющий координаты 1690.
Указанный технический результат достигается также тем, что создана полинуклеотидная вакцина, представляющая собой молекулы мРНК-С3-RBD-PolyA, индуцирующие SARS-CoV-2-специфические антитела, имеющие нуклеотидную последовательность SEQ ID NO: 6, синтезируемые плазмидной ДНК-матрицей pVAX-RBD-C3-RBD-PolyA по п. 2, полученной с использованием плазмидной ДНК-кассеты pVAX-C3-PolyA по п. 1, причем ДНК-матрица pVAX-RBD-C3-RBD-PolyA содержит открытую рамку считывания, кодирующую аминокислотную последовательность SEQ ID NO: 7, обеспечивающую синтез и секрецию белка-иммуногена RBD SARS-CoV 2 в организме млекопитающих, консенсусную последовательность Козак, 7-метилгуанозиновый кэп на 5'-конце мРНК, полиаденозиновый (поли А) хвост на 3'-конце мРНК, длиной 100 нуклеотидов, и 5'-нетранслируемую область ChM и 3'НТО β-глобина перед и после открытой рамки считывания соответственно.
Полинуклеотидная вакцина, представляющая собой молекулы мРНК-С3-RBD-PolyA, обеспечивает формирование более сильного иммунного ответа при иммунизации животных, чем при иммунизации мРНК, полученной с использованием плазмиды pVAX-RBD. Иммунизация животных осуществляется одним из способов, обеспечивающих активацию специфического гуморального и Т-клеточного иммунного ответа при введении мРНК-вакцины, например струйная инжекция или липидные наночастицы. В данной заявке на изобретение для доставки мРНК используется струйная безыгольная инжекция. Введение струйной инжекцией мРНК-вакцины, выполненной в виде молекул мРНК-С3-RBD-PolyA, индуцирующих SARS-CoV-2-специфические антитела, дважды с интервалом между вакцинациями в 3 недели в дозе 30 мкг мРНК-С3-RBD-PolyA в 50 мкл PBS с помощью струйного безыгольного инъектора с индивидуальными насадками-соплами, расположенного под прямым углом к участку поверхности для инъекции обеспечивает следующие характеристики: скорость струи 220 метров в секунду, давление 6,5 бар, время инжекции 0,33 с.
Изобретение иллюстрируется следующими графическими материалами.
На фиг. 1 представлена нуклеотидная последовательность SEQ ID NO: 1 молекул плазмидной ДНК pVAX-C3-PolyA
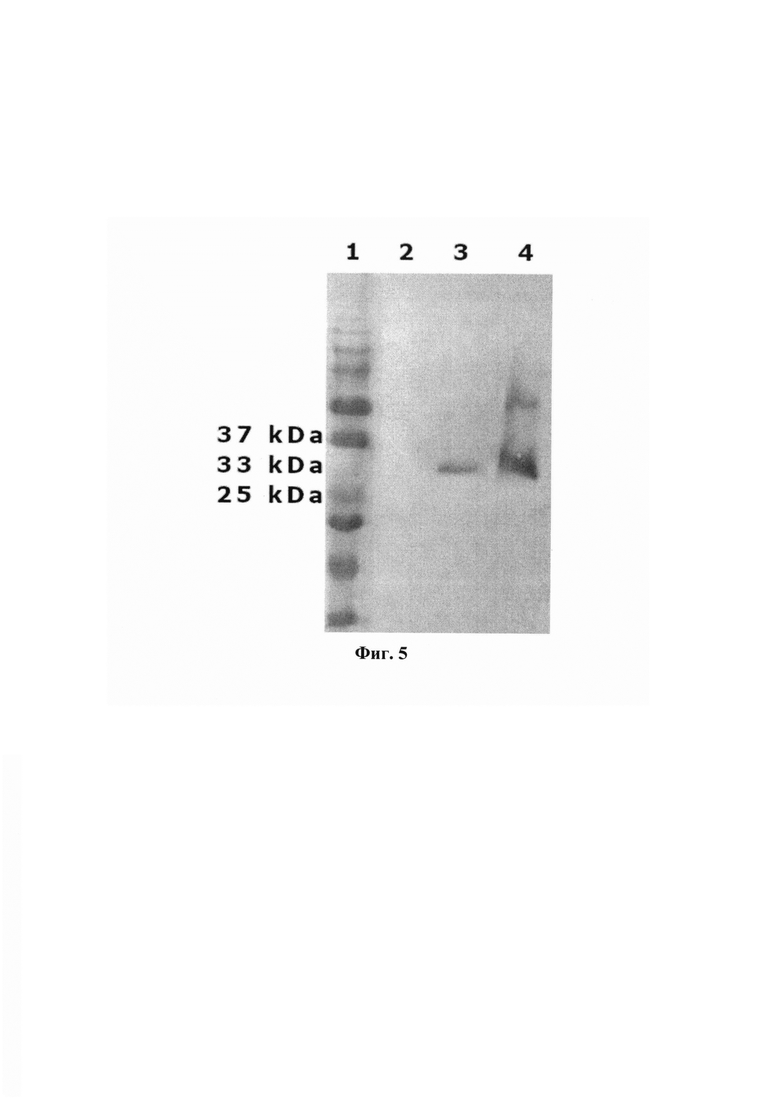
На фиг. 2 приведена нуклеотидная последовательность SEQ ID NO: 2 молекул ДНК-матрицы pVAX-C3-RBD-PolyA для синтеза мРНК; На фиг. 3 приведена нуклеотидная последовательность SEQ ID NO: 3 молекул мРНК-С3-RBD-PolyA, кодирующая последовательность белка-иммуногена 176-RBD SARS-COV-2 против SARS-COV-2. На фиг. 4 представлена аминокислотная последовательность SEQ ID NO: 4, обеспечивающая синтез и секрецию белка-иммуногена 176-RBD SARS-CoV 2 в организме млекопитающих. На фиг. 5 представлены результаты вестерн-блота культуральной среды клеток НЕК293, трансфицированных мРНК-С3-RBD-PolyA. 1 - Маркер Precision Plus Protein Dual Color Standards, 2 - культуральная среда нетрансфицированных клеток НЕК293 (отрицательный контроль), 3 - культуральная среда клеток, трансфицированных мРНК-С3-RBD-PolyA, 4 - рекомбинантный белок RBD (положительный контроль). На фиг. 6 представлен гуморальный иммунный ответ у мышей BALB/c, иммунизированных мРНК-RBD и mPHK-C3-RBD-PolyA. Титры специфических IgG антител к RBD в сыворотках иммунизированных животных к рекомбинантному белку RBD. Достоверность была рассчитана с использованием непараметрического анализа Манна-Уитни. Статистический анализ проводился с использованием программного обеспечения GraphPad Prism 8.0. (*** р<0,001, **** р<0,0001). На фиг. 7 представлены результаты анализа ELISpot специфического для SARS-CoV-2 Т-клеточного опосредованного ответа у иммунизированных мРНК-RBD и мРНК-С3-RBD-PolyA мышей BALB/c. Количество клеток, экспрессирующих IFN-γ в ответ на стимуляцию пулом RBD-специфических пептидов, на 1×106 спленоцитов. Значимость оценивали с помощью непараметрического однофакторного дисперсионного анализа Краскела-Уоллиса с поправкой на множественные сравнения и проверки статистической гипотезы Данна (** р<0,001). На фиг. 8 представлена физическая и генетическая карта плазмидной ДНК-кассеты pVAX-C3-PolyA. На фиг. 9 представлена физическая и генетическая карта плазмидной ДНК-матрицы pVAX-C3-RBD-PolyA.
Ниже приведены примеры конкретного выполнения изобретения.
Пример 1. Дизайн рекомбинантной плазмидной ДНК-кассеты pVAX-С3-PolyA, ее наработка и очистка
Дизайн плазмиды pVAX-C3-PolyA. Для конструирования ДНК-кассеты использовали вектор pVAX ("Thermo Fisher Scientific", США). В него были встроены необходимые элементы для эффективной экспрессии желаемой мРНК-вакцины. Для одностадийного синтеза мРНК необходимо использовать котранскрипционное кэпирование химическим аналогом кэпа, например AG-cap аналог (m7GmAmG), и встроить последовательность поли(А)хвоста непосредственно в ДНК-матрицу. Для повышения уровня экспрессии мРНК необходимо использование НТО из высокоэкспрессируемых генов, так как они повышают уровень экспрессии и стабильность РНК.
Для использования AG-Cap аналога было произведено изменение Т7 промотора, инициирующие кодоны GGG были заменены на AGG.
В качестве 5'НТО была использована модифицированная химерная последовательность, предложенная компанией Moderna для мРНК-вакцины против COVID-19 (ChM), а в качестве 3'НТО последовательность β-глобина человека. Последовательность 5'НТО заканчивалась сайтом эндонуклеазы рестрикции CciNI, а 3'НТО начиналась сайтом эндонуклеазы рестрикции BamHI. Данные сайты будут использоваться для встройки желаемого гена в ДНК-кассету.
Для полиаденилирования мРНК непосредственно при синтезе, в конструкцию был встроен поли(А)хвост длиной 100 нуклеотидов с включением внутреннего линкера из 10 случайных нуклеотидов, например 30(A)GCATATGACT70(A). Данный линкер необходим для повышения стабильности плазмиды при репликации в бактериальных клетках. После поли(А) последовательности был встроен сайт эндонуклеазы рестрикции Bso31I, обеспечивающий гидролиз ДНК сбоку от сайта узнавания, непосредственно по поли(А) хвосту, чтобы транскрипт заканчивался аденином. Это необходимо для нормального функционирования поли(А)хвоста в мРНК.
Полученную нуклеотидную последовательность синтезировали в ООО "ДНК-синтез" (Россия) и клонировали в составе вектора pVAX по сайтам Psp124BI и PspOMI. Структуру полученной конструкции подтверждали секвенированием по методу Сэнгера в ЦКП "Геномика" (Россия). В результате была получена ДНК-конструкция pVAX-C3-PolyA.
Наработка и очистка рекомбинантной плазмиды pVAX-C3-PolyA. Клетки Escherichia coli Stb13 трансформировали плазмидами pVAX и pVAX-С3-PolyA с помощью хлористого кальция. Наработку плазмидной ДНК для иммунизации проводили в 2.7 л питательной среды LB с добавлением канамицина (25 мкг/мл). Плазмидную ДНК выделяли и очищали с помощью набора EndoFree Plasmid Giga Kit ("QIAGEN", Германия) согласно рекомендациям производителя. Данный набор позволяет очень эффективно очистить препарат плазмидной ДНК от бактериальных эндотоксинов. После очистки структуру плазмиды проверяли рестрикционным анализом и спектрофотометрией.
В результате, были получены молекулы плазмидной ДНК-кассеты pVAX-C3-PolyA, имеющие нуклеотидную последовательность SEQ ID NO: 1 (фиг. 1), содержащую кассету, предназначенную для встройки в нее целевого гена для мРНК-вакцины.
Пример 2. Дизайн плазмидной ДНК-матрицы pVAX-C3-RBD-PolyA для синтеза мРНК
Для получения ДНК-матрицы использовали вектор pVAX-C3-PolyA. В него встраивали последовательность белка RBD по сайтам CciNI и BamHI, в результате получили вектор pVAX-C3-RBD-PolyA.
Для конструирования иммуногена использовали последовательность гена, кодирующего полноразмерный белок S SARS-CoV-2 (GenBank Acc. No. MN908947). Для оптимизации состава кодонов, а также вторичной структуры РНК для эффективной экспрессии в клетках млекопитающих использовали программу GeneOptimizer (https://www.thermofisher.com/ru/en/home/life-science/cloning/gene-synthesis/geneart-gene-synthesis/geneoptimizer.html).
Полученную нуклеотидную последовательность синтезировали в ООО "ДНК-синтез" (Россия) и клонировали в составе вектора. Для амплификации фрагмента, кодирующего RBD белка S (320V-542N) и сигнальную последовательность 176 (MMRTLILAVLLVYFCATVHC) на 5'-конце, использовали праймеры: 5'- GCGGCCGCCACCATGATGCGGACACTGATC-3' (прямой) и 5'- GGATCCTTAGTTGAAGTTCACGCATTT-3' (обратный), - и матрицу, содержащую указанный фрагмент.
Продукт амплификации встраивали в вектор pVAX-C3-PolyA по сайтам CciNI и BamHI. Структуру полученной конструкции подтверждали секвенированием по методу Сэнгера в ЦКП "Геномика" (Россия). В результате была получена ДНК-конструкция pVAX-C3-RBD-PolyA, кодирующая RBD с сигнальной последовательностью 176, обеспечивающей секрецию нарабатываемого белка из клетки. Данная конструкция является ДНК-матрицей для синтеза мРНК.
Пример 3. Получение молекул мРНК-С3-RBD-PolyA
Синтез мРНК кодирующей RBD SARS-COV-2 проводили с линеаризованной и очищенной ДНК-матрицы pVAX-C3-RBD-PolyA, имеющей нуклеотидную последовательность SEQ ID NO: 2 (фиг. 2), и содержащей промотор РНК-полимеразы бактериофага Т7, 5'НТО, целевой ген GFP, 3'НТО и поли(А)хвост длиной 100 нуклеотидов.
Реакцию транскрипции проводили с помощью РНК-полимеразы бактериофага Т7 (БиоЛабМикс, Новосибирск, Россия). Реакционная смесь включала в себя 1 мкг линеаризованной матрицы, Т7 полимеразу с буфером, смесь рибонуклеотидов, в которой уридин был заменен на псевдоуридин или метилпсевдоуридин, аналог структуры кэпа m7GmAmG (БиоЛабМикс, Новосибирск, Россия), ингибитор РНКаз и безнуклеазную воду. Смесь инкубировали в течение 2 часов при температуре 37°С. После инкубации в смесь добавляли 1 мкл ДНКазы, и инкубировали в течение 30 минут при температуре 37°С для разрушения ДНК матрицы. Анализ полученного продукта производили с использованием электрофореза в 2% агарозном геле.
В результате была получена мРНК, имеющая нуклеотидную последовательность SEQ ID NO: 3 (фиг. 3), которая была способна обеспечить синтез в эукариотической клетке целевого белка RBD SARS-COV-2 с аминокислотной последовательностью SEQ ID NO: 4 (фиг. 4).
В качестве контроля с плазмиды pVAX-RBD была получена mPHK-RBD. Данная плазмида имеет обычный Т7 промотор, и не имеет последовательностей НТО и поли(А)хвоста. Поэтому кэпирование и полиаденилирование РНК проводили коммерческими ферментами.
Пример 4. Анализ экспрессии мРНК-C1-RBD в трансфицированных клетках HEK-293
Для анализа работоспособности мРНК проводили трансфекцию культуры клеток HEK293 мРНК-С3-RBD-PolyA с помощью Lipofectamine MessengerMAX. Вестерн-блот культуральной среды трансфицированных клеток с мышиной сывороткой к RBD подтвердил, что мРНК-С3-RBD-PolyA обеспечивает синтез целевого вирусного белка в эукариотических клетках (фиг. 5).
Пример 5. Доставка экспериментальной мРНК-вакцины, кодирующей RBD SARS-CoV-2 с помощью струйной инжекции
Для исследования иммуногенности мРНК-вакцины были использованы мыши линии Balb/c (самки) массой 16-18 г. Эксперименты с животными проводили с соблюдением принципов гуманности в соответствии с протоколами, утвержденными Биоэтическим комитетом ГНЦ ВБ «Вектор»/02-03.2023, Протокол БЭК №2 от 03.04.2023 г.). Было сформировано 3 группы по 8 животных в каждой. Волосы удаляли с волосистой части лапы с помощью геля для депиляции. Иммунизацию мышей проводили в четырехглавую мышцу задней левой лапы дважды на 0 и 21 дни с помощью струйной инжекции. Первой группе животных вводили 30 мкг мРНК-RBD в 50 мкл PBS; второй группе - в/м 30 мкг мРНК-С3-RBD-PolyA в 50 мкл PBS; третьей группе вводили в/м 50 мкл физиологического раствора. Струйную инжекцию проводили с использованием собственного опытного образца инжектора с характеристиками: скорость струи 220 метров в секунду, давление 6,5 бар, время инжекции 0,33 с. используя индивидуальные насадки-сопло. На 10-й день после второй инъекции у мышей брали кровь, умерщвляли и собирали их кровь и селезенки для дальнейшего анализа иммуногенности.
ИФА сыворотки. В качестве антигенов для ELISA использовали эукариотические белки RBD, любезно предоставленные лабораторией иммунохимии ФБУН ГНЦ ВБ Вектор Роспотребнадзора (Россия). Белок RBD (1 мкг / мл) адсорбировали в лунках 96-луночного планшета в PBS (Greiner_bio one, Германия) при 4°С в течение 12 часов, затем промывали PBST и блокировали с 1% раствором казеина в промывочном буфере в течение 60 минут при комнатной температуре. После этого образцы сыворотки добавляли в трехкратном серийном разведении, начиная с 1:50, и инкубировали в течение 60 минут при 37°С. После промывания добавляли кроличьи антитела против мышиного IgG, конъюгированные с пероксидазой хрена (Sigma, США), и инкубировали в течение 60 минут при 37°С. Планшет промывали и добавляли к нему раствор субстрата ТМВ (Amresco, США). После остановки реакции стоп-раствором оптическую плотность измеряли при длине волны 450 нм с помощью считывающего устройства для ELISA (ChroMate Awareness Technology inc., США).
Анализ IFN-γ ELISpot. Анализ проводился с использованием наборов Mabtech (США, Цинциннати) в соответствии с рекомендациями производителя. Спленоциты, выделенные от иммунизированных животных, стимулировали пулом из 13 RBD-пептидов SARS-CoV-2 (по 20 мкг/мл каждого пептида), распознаваемых главным комплексом гистосовместимости (МНС) класса I (H-2-Dd, H-2-Kd, H-2-Ld) и молекулы класса II (H2-IAd, Н2-IEd) для мышей BALB/c. Пептиды синтезированы фирмой AtaGenix Laboratories (Китай, г. Ухань), чистота пептидов > 80%. Количество клеток, продуцирующих ИФН-γ, подсчитывали с помощью анализатора ELISpot фирмы Carl Zeiss (Германия, г. Оберкохен).
Статистика. Статистическую обработку полученных данных проводили с использованием непараметрического анализа Манна-Уитни в программе GraphPad Prism 9.0, при р<0.05 различия считали статистически значимыми.
Для анализа иммуногенности была проведена иммунизация лабораторных животных, которым вводили по 30 мкг мРНК-С3-RBD с помощью струйной инжекции, а в качестве группы сравнения использовали животных, иммунизированных мРНК-RBD. ИФА анализ сывороток животных, показал, что титр RBD-специфических антител у мышей, иммунизированных мРНК, полученной с разработанной кассеты pVAX-C3-RBD-PolyA, мРНК-С3-RBD-PolyA, значительно превышают титр антител у мышей контрольной группы (фиг. 6).
Клеточный ответ анализировали с помощью метода ELISpot. Было показано количество спленоцитов, продуцирующих IFN-γ в ответ на стимуляцию RBD-специфическими пептидами, в группе мРНК-С3-RBD-PolyA, превышал таковой в группе mPHK-RBD (фиг. 7).
В контрольных группах животных, иммунизированных физиологическим раствором, клеточный ответ был на уровне фона. Эти данные свидетельствуют об увеличении специфического поствакцинального Т-клеточного ответа при использовании струйной инжекции, что коррелирует с усилением гуморального иммунного ответа.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="Приложение.
Плазмидная ДНК-кассета, плазмидная ДНК-матрица для синтеза мРНК и
полинуклеотидная вакцина..xml" softwareName="WIPO Sequence"
softwareVersion="2.3.0" productionDate="2024-10-28">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>123456</ApplicationNumberText>
<FilingDate>2024-10-28</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>123</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>1234567</ApplicationNumberText>
<FilingDate>2024-10-19</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">Федеральное бюджетное учреждение
науки «Государственный научный центр вирусологии и биотехнологии
«Вектор» Федеральной службы по надзору в сфере защиты прав
потребителей и благополучия человека (ФБУН ГНЦ ВБ «Вектор»
Роспотребнадзора)</ApplicantName>
<ApplicantNameLatin>Federalnoe byudzhetnoe uchrezhdenie nauki
"Gosudarstvennyj nauchnyj tsentr virusologii i biotekhnologii
"Vektor" Federalnoj sluzhby po nadzoru v sfere zashchity
prav potrebitelej i blagopoluchiya cheloveka (FBUN GNTS VB
"Vektor" Rospotrebnadzora) (RU)</ApplicantNameLatin>
<InventionTitle languageCode="ru">Плазмидная ДНК-кассета
pVAX-C3-PolyA, плазмидная ДНК-матрица pVAX-С3-RBD-PolyA для синтеза
мРНК и полинуклеотидная вакцина, представляющая собой молекулы
мРНК-С3-RBD-PolyA, индуцирующие SARS-CoV-2-специфические
антитела</InventionTitle>
<SequenceTotalQuantity>4</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>3171</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..3171</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gactcttcgcgatgtacgggccagatatacgcgttgacattgattattg
actagttattaatagtaatcaattacggggtcattagttcatagcccatatatggagttccgcgttacat
aacttacggtaaatggcccgcctggctgaccgcccaacgacccccgcccattgacgtcaataatgacgta
tgttcccatagtaacgccaatagggactttccattgacgtcaatgggtggactatttacggtaaactgcc
cacttggcagtacatcaagtgtatcatatgccaagtacgccccctattgacgtcaatgacggtaaatggc
ccgcctggcattatgcccagtacatgaccttatgggactttcctacttggcagtacatctacgtattagt
catcgctattaccatggtgatgcggttttggcagtacatcaatgggcgtggatagcggtttgactcacgg
ggatttccaagtctccaccccattgacgtcaatgggagtttgttttggcaccaaaatcaacgggactttc
caaaatgtcgtaacaactccgccccattgacgcaaatgggcggtaggcgtgtacggtgggaggtctatat
aagcagagctcaaattaatacgactcactataagggaaataagagagaaaagaagagtaagaagaaatat
aagaccccggcggccgccaccggatcctaagctcgctttcttgctgtccaatttctattaaaggttcctt
tgttccctaagtccaactactaaactgggggatattatgaagggccttgagcatctggattctgcctaat
aaaaaacatttattttcattgcgaattcaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaagcatatgacta
aaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaa
aaaagagaccgggcccgtttaaacccgctgatcagcctcgactgtgccttctagttgccagccatctgtt
gtttgcccctcccccgtgccttccttgaccctggaaggtgccactcccactgtcctttcctaataaaatg
aggaaattgcatcgcattgtctgagtaggtgtcattctattctggggggtggggtggggcaggacagcaa
gggggaggattgggaagacaatagcaggcatgctggggatgcggtgggctctatggcttctactgggcgg
ttttatggacagcaagcgaaccggaattgccagctggggcgccctctggtaaggttgggaagccctgcaa
agtaaactggatggctttctcgccgccaaggatctgatggcgcaggggatcaagctctgatcaagagaca
ggatgaggatcgtttcgcatgattgaacaagatggattgcacgcaggttctccggccgcttgggtggaga
ggctattcggctatgactgggcacaacagacaatcggctgctctgatgccgccgtgttccggctgtcagc
gcaggggcgcccggttctttttgtcaagaccgacctgtccggtgccctgaatgaactgcaagacgaggca
gcgcggctatcgtggctggccacgacgggcgttccttgcgcagctgtgctcgacgttgtcactgaagcgg
gaagggactggctgctattgggcgaagtgccggggcaggatctcctgtcatctcaccttgctcctgccga
gaaagtatccatcatggctgatgcaatgcggcggctgcatacgcttgatccggctacctgcccattcgac
caccaagcgaaacatcgcatcgagcgagcacgtactcggatggaagccggtcttgtcgatcaggatgatc
tggacgaagagcatcaggggctcgcgccagccgaactgttcgccaggctcaaggcgagcatgcccgacgg
cgaggatctcgtcgtgacccatggcgatgcctgcttgccgaatatcatggtggaaaatggccgcttttct
ggattcatcgactgtggccggctgggtgtggcggaccgctatcaggacatagcgttggctacccgtgata
ttgctgaagagcttggcggcgaatgggctgaccgcttcctcgtgctttacggtatcgccgctcccgattc
gcagcgcatcgccttctatcgccttcttgacgagttcttctgaattattaacgcttacaatttcctgatg
cggtattttctccttacgcatctgtgcggtatttcacaccgcatacaggtggcacttttcggggaaatgt
gcgcggaacccctatttgtttatttttctaaatacattcaaatatgtatccgctcatgagacaataaccc
tgataaatgcttcaataatagcacgtgctaaaacttcatttttaatttaaaaggatctaggtgaagatcc
tttttgataatctcatgaccaaaatcccttaacgtgagttttcgttccactgagcgtcagaccccgtaga
aaagatcaaaggatcttcttgagatcctttttttctgcgcgtaatctgctgcttgcaaacaaaaaaacca
ccgctaccagcggtggtttgtttgccggatcaagagctaccaactctttttccgaaggtaactggcttca
gcagagcgcagataccaaatactgtccttctagtgtagccgtagttaggccaccacttcaagaactctgt
agcaccgcctacatacctcgctctgctaatcctgttaccagtggctgctgccagtggcgataagtcgtgt
cttaccgggttggactcaagacgatagttaccggataaggcgcagcggtcgggctgaacggggggttcgt
gcacacagcccagcttggagcgaacgacctacaccgaactgagatacctacagcgtgagctatgagaaag
cgccacgcttcccgaagggagaaaggcggacaggtatccggtaagcggcagggtcggaacaggagagcgc
acgagggagcttccagggggaaacgcctggtatctttatagtcctgtcgggtttcgccacctctgacttg
agcgtcgatttttgtgatgctcgtcaggggggcggagcctatggaaaaacgccagcaacgcggccttttt
acggttcctgggcttttgctggccttttgctcacatgttctt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>3903</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..3903</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gactcttcgcgatgtacgggccagatatacgcgttgacattgattattg
actagttattaatagtaatcaattacggggtcattagttcatagcccatatatggagttccgcgttacat
aacttacggtaaatggcccgcctggctgaccgcccaacgacccccgcccattgacgtcaataatgacgta
tgttcccatagtaacgccaatagggactttccattgacgtcaatgggtggactatttacggtaaactgcc
cacttggcagtacatcaagtgtatcatatgccaagtacgccccctattgacgtcaatgacggtaaatggc
ccgcctggcattatgcccagtacatgaccttatgggactttcctacttggcagtacatctacgtattagt
catcgctattaccatggtgatgcggttttggcagtacatcaatgggcgtggatagcggtttgactcacgg
ggatttccaagtctccaccccattgacgtcaatgggagtttgttttggcaccaaaatcaacgggactttc
caaaatgtcgtaacaactccgccccattgacgcaaatgggcggtaggcgtgtacggtgggaggtctatat
aagcagagctcaaattaatacgactcactataagggaaataagagagaaaagaagagtaagaagaaatat
aagaccccggcggccgccaccatgatgcggacactgatcctggctgtgctgctggtgtacttctgtgcca
ccgtgcactgtgtgcagcccaccgaatccatcgtgcggttccccaatatcaccaatctgtgccccttcgg
cgaggtgttcaatgccaccagattcgcctctgtgtacgcctggaaccggaagcggatcagcaattgcgtg
gccgactactccgtgctgtacaactccgccagcttcagcaccttcaagtgctacggcgtgtcccctacca
agctgaacgacctgtgcttcacaaacgtgtacgccgacagcttcgtgatccggggagatgaagtgcggca
gattgcccctggacagacaggcaagatcgccgactacaactacaagctgcccgacgacttcaccggctgt
gtgattgcctggaacagcaacaacctggactccaaagtcggcggcaactacaattacctgtaccggctgt
tccggaagtccaatctgaagcccttcgagcgggacatctccaccgagatctatcaggccggcagcacccc
ttgtaacggcgtggaaggcttcaactgctacttcccactgcagtcctacggctttcagcccacaaatggc
gtgggctatcagccctacagagtggtggtgctgagcttcgaactgctgcatgcccctgccacagtgtgcg
gccctaagaaaagcaccaatctcgtgaagaacaaatgcgtgaacttcaactaaggatcctaagctcgctt
tcttgctgtccaatttctattaaaggttcctttgttccctaagtccaactactaaactgggggatattat
gaagggccttgagcatctggattctgcctaataaaaaacatttattttcattgcgaattcaaaaaaaaaa
aaaaaaaaaaaaaaaaaaaaagcatatgactaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaa
aaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaagagaccgggcccgtttaaacccgctgatcagcct
cgactgtgccttctagttgccagccatctgttgtttgcccctcccccgtgccttccttgaccctggaagg
tgccactcccactgtcctttcctaataaaatgaggaaattgcatcgcattgtctgagtaggtgtcattct
attctggggggtggggtggggcaggacagcaagggggaggattgggaagacaatagcaggcatgctgggg
atgcggtgggctctatggcttctactgggcggttttatggacagcaagcgaaccggaattgccagctggg
gcgccctctggtaaggttgggaagccctgcaaagtaaactggatggctttctcgccgccaaggatctgat
ggcgcaggggatcaagctctgatcaagagacaggatgaggatcgtttcgcatgattgaacaagatggatt
gcacgcaggttctccggccgcttgggtggagaggctattcggctatgactgggcacaacagacaatcggc
tgctctgatgccgccgtgttccggctgtcagcgcaggggcgcccggttctttttgtcaagaccgacctgt
ccggtgccctgaatgaactgcaagacgaggcagcgcggctatcgtggctggccacgacgggcgttccttg
cgcagctgtgctcgacgttgtcactgaagcgggaagggactggctgctattgggcgaagtgccggggcag
gatctcctgtcatctcaccttgctcctgccgagaaagtatccatcatggctgatgcaatgcggcggctgc
atacgcttgatccggctacctgcccattcgaccaccaagcgaaacatcgcatcgagcgagcacgtactcg
gatggaagccggtcttgtcgatcaggatgatctggacgaagagcatcaggggctcgcgccagccgaactg
ttcgccaggctcaaggcgagcatgcccgacggcgaggatctcgtcgtgacccatggcgatgcctgcttgc
cgaatatcatggtggaaaatggccgcttttctggattcatcgactgtggccggctgggtgtggcggaccg
ctatcaggacatagcgttggctacccgtgatattgctgaagagcttggcggcgaatgggctgaccgcttc
ctcgtgctttacggtatcgccgctcccgattcgcagcgcatcgccttctatcgccttcttgacgagttct
tctgaattattaacgcttacaatttcctgatgcggtattttctccttacgcatctgtgcggtatttcaca
ccgcatacaggtggcacttttcggggaaatgtgcgcggaacccctatttgtttatttttctaaatacatt
caaatatgtatccgctcatgagacaataaccctgataaatgcttcaataatagcacgtgctaaaacttca
tttttaatttaaaaggatctaggtgaagatcctttttgataatctcatgaccaaaatcccttaacgtgag
ttttcgttccactgagcgtcagaccccgtagaaaagatcaaaggatcttcttgagatcctttttttctgc
gcgtaatctgctgcttgcaaacaaaaaaaccaccgctaccagcggtggtttgtttgccggatcaagagct
accaactctttttccgaaggtaactggcttcagcagagcgcagataccaaatactgtccttctagtgtag
ccgtagttaggccaccacttcaagaactctgtagcaccgcctacatacctcgctctgctaatcctgttac
cagtggctgctgccagtggcgataagtcgtgtcttaccgggttggactcaagacgatagttaccggataa
ggcgcagcggtcgggctgaacggggggttcgtgcacacagcccagcttggagcgaacgacctacaccgaa
ctgagatacctacagcgtgagctatgagaaagcgccacgcttcccgaagggagaaaggcggacaggtatc
cggtaagcggcagggtcggaacaggagagcgcacgagggagcttccagggggaaacgcctggtatcttta
tagtcctgtcgggtttcgccacctctgacttgagcgtcgatttttgtgatgctcgtcaggggggcggagc
ctatggaaaaacgccagcaacgcggcctttttacggttcctgggcttttgctggccttttgctcacatgt
tctt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>1049</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..1049</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agggaaataagagagaaaagaagagtaagaagaaatataagaccccggc
ggccgccaccatgatgcggacactgatcctggctgtgctgctggtgtacttctgtgccaccgtgcactgt
gtgcagcccaccgaatccatcgtgcggttccccaatatcaccaatctgtgccccttcggcgaggtgttca
atgccaccagattcgcctctgtgtacgcctggaaccggaagcggatcagcaattgcgtggccgactactc
cgtgctgtacaactccgccagcttcagcaccttcaagtgctacggcgtgtcccctaccaagctgaacgac
ctgtgcttcacaaacgtgtacgccgacagcttcgtgatccggggagatgaagtgcggcagattgcccctg
gacagacaggcaagatcgccgactacaactacaagctgcccgacgacttcaccggctgtgtgattgcctg
gaacagcaacaacctggactccaaagtcggcggcaactacaattacctgtaccggctgttccggaagtcc
aatctgaagcccttcgagcgggacatctccaccgagatctatcaggccggcagcaccccttgtaacggcg
tggaaggcttcaactgctacttcccactgcagtcctacggctttcagcccacaaatggcgtgggctatca
gccctacagagtggtggtgctgagcttcgaactgctgcatgcccctgccacagtgtgcggccctaagaaa
agcaccaatctcgtgaagaacaaatgcgtgaacttcaactaaggatcctaagctcgctttcttgctgtcc
aatttctattaaaggttcctttgttccctaagtccaactactaaactgggggatattatgaagggccttg
agcatctggattctgcctaataaaaaacatttattttcattgcgaattcaaaaaaaaaaaaaaaaaaaaa
aaaaaaaaaagcatatgactaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaa
aaaaaaaaaaaaaaaaaaaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>243</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..243</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MMRTLILAVLLVYFCATVHCVQPTESIVRFPNITNLCPFGEVFNATRFA
SVYAWNRKRISNCVADYSVLYNSASFSTFKCYGVSPTKLNDLCFTNVYADSFVIRGDEVRQIAPGQTGKI
ADYNYKLPDDFTGCVIAWNSNNLDSKVGGNYNYLYRLFRKSNLKPFERDISTEIYQAGSTPCNGVEGFNC
YFPLQSYGFQPTNGVGYQPYRVVVLSFELLHAPATVCGPKKSTNLVKNKCVNFN</INSDSeq_sequen
ce>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---








Изобретение относится к биотехнологии. Создана плазмидная ДНК-кассета pVAX-C3-PolyA, используемая для встройки в нее целевого гена для формирования матрицы для синтеза мРНК, имеющая нуклеотидную последовательность SEQ ID NO: 1 размером 3171 п.н., молекулярный вес 1,9 МДа. Создана плазмидная ДНК-матрица pVAX-C3-PvBD-PolyA для синтеза мРНК, полученная с использованием плазмидной ДНК-кассеты pVAX-C3-PolyA по п. 1, имеющая нуклеотидную последовательность SEQ ID NO: 2 размером 3903 п.н., молекулярный вес 2,3 МДа, включающая целевой ген, кодирующий химерный белок 176-RBD, имеющий аминокислотную последовательность SEQ ID NO: 4 и координаты 701 по 1432. Получена полинуклеотидная вакцина, представляющая собой молекулы мРНК-С3-RBD-PolyA, индуцирующие SARS-CoV-2-специфические антитела, имеющие нуклеотидную последовательность SEQ ID NO: 3, синтезируемые плазмидной ДНК-матрицей pVAX-RBD-C3-RBD-PolyA по п. 2, полученной с использованием плазмидной ДНК-кассеты pVAX-C3-PolyA по п. 1. Техническим результатом заявляемого изобретения является разработка платформы в виде ДНК-кассеты и ДНК-матрицы на ее основе для синтеза мРНК. 3 н.п. ф-лы, 9 ил., 5 пр.
1. Плазмидная ДНК-кассета pVAX-C3-PolyA, используемая для встройки в нее целевого гена для формирования матрицы для синтеза мРНК, имеющая нуклеотидную последовательность SEQ ID NO: 1 размером 3171 п.н., молекулярный вес 1,9 МДа, и содержащая следующие элементы конструкции:
- векторный фрагмент ДНК-плазмиды pVAX, имеющий размер 2837 п.н. и координаты 958-624;
- ген устойчивости к неомицину/канамицину (NeoR/KanR) с координатами 1398-2192 размером 795 п.н. и точка начала репликации ColEl ori с координатами 2518-3106 и размером 589 п.н., обеспечивающие селекцию и амплификацию целевой плазмиды в клетках бактерий Escherichia coli;
- фрагмент размером 20 п.н., имеющий координаты 625-644, являющийся промотором РНК-полимеразы фага Т7, с инициирующими нуклеотидами AGG и необходимый для использования AG-Cap аналога;
- фрагмент размером 56 п.н., имеющий координаты 645-700, являющийся модифицированной химерной последовательностью (ChM) и используемый в качестве 5'НТО для мРНК-вакцины против COVID-19;
- фрагмент размером 135 п.н., имеющий координаты 707-841, являющийся последовательностью β-глобина человека и используемый в качестве 3'НТО;
- фрагмент размером 111 п.н., имеющий координаты 848-958, представляющий собой поли(А)хвост длиной 100 нуклеотидов с внутренним линкером из 10 нуклеотидов 30(A)GCATATGACT70(A);
- сайты эндонуклеазы рестрикции CciNI и BamHI, имеющие координаты 690 и 701 соответственно;
- сайт эндонуклеазы рестрикции Bso31I, имеющий координаты 958.
2. Плазмидная ДНК-матрица pVAX-C3-RBD-PolyA для синтеза мРНК, полученная с использованием плазмидной ДНК-кассеты pVAX-C3-PolyA по п. 1, имеющая нуклеотидную последовательность SEQ ID NO: 2 размером 3903 п.н., молекулярный вес 2,3 МДа, включающая целевой ген, кодирующий химерный белок 176-RBD, имеющий аминокислотную последовательность SEQ ID NO: 4 и координаты 701 по 1432, и состоящая из следующих фрагментов:
- векторный фрагмент ДНК-плазмиды pVAX, имеющий размер 2837 п.н. и координаты 1690-624;
- ген устойчивости к неомицину/канамицину (NeoR/KanR), имеющий координаты 2130-2924 и размер 795 п.н., и точка начала репликации ColEl ori, имеющая координаты 3250-3838 и размер 589 п.н., обеспечивающие селекцию и амплификацию целевой плазмиды в клетках бактерий Escherichia coli;
- фрагмент размером 20 п.н., имеющий координаты 625-644 и являющийся промотором РНК-полимеразы фага Т7 с инициирующими нуклеотидами AGG, обеспечивающий транскрипцию гена, кодирующего химерный белок 176-RBD;
- фрагмент размером 56 п.н., имеющий координаты 645-700, являющийся модифицированной химерной последовательностью (ChM) и используемый в качестве 5'НТО для мРНК-вакцины против COVID-19;
- фрагмент размером 732 п.н., имеющий координаты 701-1432, содержащий искусственный ген 176-RBD с инициирующим кодоном ATG;
- фрагмент размером 135 п.н., имеющий координаты 1439-1573, являющийся последовательностью β-глобина человека и используемый в качестве 3'НТО;
- фрагмент размером 111 п.н., имеющий координаты 1580-1690, представляющий собой поли(А)хвост длиной 100 нуклеотидов с внутренним линкером из 10 нуклеотидов 30(A)GCATATGACT70(A);
- сайты эндонуклеазы рестрикции CciNI и BamHI, имеющие координаты 690 и 1433 соответственно;
- сайт эндонуклеазы рестрикции Bso31I, имеющий координаты 1690.
3. Полинуклеотидная вакцина для профилактики коронавирусной инфекции, представляющая собой молекулы мРНК-С3-RBD-PolyA, индуцирующие SARS-CoV-2-специфические антитела, имеющие нуклеотидную последовательность SEQ ID NO: 3, синтезируемые плазмидной ДНК-матрицей pVAX-RBD-C3-RBD-PolyA по п. 2, полученной с использованием плазмидной ДНК-кассеты pVAX-C3-PolyA по п. 1, причем ДНК-матрица pVAX-RBD-C3-RBD-PolyA содержит открытую рамку считывания, кодирующую аминокислотную последовательность SEQ ID NO: 4, обеспечивающую синтез и секрецию белка-иммуногена RBD SARS-CoV 2 в организме млекопитающих, консенсусную последовательность Козак, 7-метилгуанозиновый кэп на 5'-конце мРНК, полиаденозиновый (поли А) хвост на 3'-конце мРНК, длиной 100 нуклеотидов, и 5'-нетранслируемую область ChM, и 3'НТО β-глобина перед и после открытой рамки считывания соответственно.
Авторы
Даты
2025-05-13—Публикация
2024-11-14—Подача