Область техники, к которой относится изобретение
Изобретение относится к области биотехнологии и генной инженерии растений, а именно к генетическим маркерам, выявляющим редактирование гена ISA1 в геноме твердой пшеницы (Triticum durum) и озимой и яровой тритикале (× Triticosecale).
Предшествующий уровень техники
Крахмал, содержащийся в эндосперме зерна, играет ключевую роль в определении качества зерна, включая его вкусовые характеристики и пищевую ценность. Мутация генов пути биосинтеза крахмала оказывает влияние на качество и количество крахмала и является важной целью для селекции растений (Chen et al., 2021). Крахмал состоит из двух полимеров глюкозы: амилозы, образованной линейными глюкозидными цепями, и амилопектина. Амилопектин основной компонент крахмала (65-85%) образующий кластерные структуры (Thompson, 2000). Известно, что в биосинтезе амилозы и амилопектина участвует широкий спектр ферментов, например, фермент разветвления крахмала DBE (Pfister В. and Zeeman S.C., 2016). Ранее в исследованиях были идентифицированы два типа DBE: изоамилаза (ISA1, ISA2 и ISA3) и пуллуланаза (PUL) (Kubo et al., 2005). Исследования показали, что мутации в гене ISA1 приводят к значительному изменению структуры амилопектина (Ferreira et al., 2017). Нокаут гена ISA1 в твердой пшенице изменяет состав крахмала в эндосперме, что приводит к незначительному снижению содержания крахмала, на фоне значительного улучшения его качества (Sestili et al, 2016). Вот почему для ускорения процесса селекции твердой пшеницы и тритикале, мы использовали систему редактирования генома с применением CRISPR/Cas9, направленную на изменение гена ISA1.
Редактирование с помощью системы CRISPR/Cas9 - высокоточный молекулярный метод, способный внести точечный разрыв в необходимое место генома, меченное с помощью специально подобранной гидовой РНК, комплементарной затравки - мишени для Cas9. Впервые система редактирования с помощью CRISPR/Cas9 была использована для редактирования генома растений еще в 2013 году (Wenzhi J. at al., 2013). В ходе развития технологии редактирование гена ISA1 проводилось разными способами на таких культурах, как арабидопсис (Wattebled et al., 2005), кукуруза (James et al., 1995), рис (Kubo et al., 1999), ячмень (Burton et al., 2002), картофель (Ferreira et al., 2017) и наконец пшеница (Sestili et al., 2016).
После проведения редактирования необходимо подтвердить наличие мутаций в каждом из сайтов редактирования для дальнейшей дифференциации растений на редактированные и нередактированные, во всех вышеперечисленных работах генетические модификации выявляли с помощью секвенирования следующего поколения (NGS).
Секвенирование следующего поколения (NGS) представляет собой передовую биологическую технологию, используемую для определения последовательности нуклеотидов в геноме с высокой точностью и эффективностью. Подготовительным этапом для проведения секвенирования является подготовка генетического материала, включающая фрагментацию и прикрепление адаптеров, амплификацию образцов и создание кластеров на поверхности чипа или потока. Далее следует этап секвенирования, в процессе которого получают короткие последовательности, называемые "ридами". Риды обрабатываются и выравниваются на референсный геном или собираются в контиги с использованием биоинформатических инструментов.
Секвенирование нового поколения (NGS) представляет собой мощный инструмент для исследования генетической информации, но оно также является трудоемким и дорогостоящим процессом по нескольким факторам. Во-первых, NGS генерирует огромные объемы данных, особенно при анализе целых геномов или больших наборов образцов, что требует значительных вычислительных ресурсов и времени для их обработки и анализа. Во-вторых, секвенирование требует использования дорогостоящего оборудования и химических реагентов, что увеличивает финансовые затраты на проведение исследований. В-третьих, эффективное использование NGS требует высокой квалификации и опыта в обработке данных и биоинформатике, что делает этот процесс трудоемким и зависящим от наличия опытного исполнителя. Наконец, анализ и интерпретация данных, полученных с помощью NGS, также являются сложными и времязатратными процессами.
Проведение каждой новой модификации представляет собой индивидуальный процесс, который обусловлен необходимостью подбора новых сайтов и гидовых последовательностей для проведения редактирования. В связи с этим каждое новое редактирование требует создания уникальных олигонуклеотидных праймеров, которые фланкируют область редактирования и специфичны для данного эксперимента. Олигонуклеотиды, использованные ранее в аналогичных экспериментах, не подходят для выявления проведенного нами редактирования, так как в нашем случае использовались новые сайты редактирования, и их количество также отличалось от предыдущих работ.
Таким образом, существует потребность более простом, способе обнаружения изменений нуклеотидных последовательностей целевых генов, в частности гена ISA1, возникающих в геноме в результате редактирования с помощью системы CRISPR/Cas9. Настоящее изобретение предлагает такой способ.
Сущность изобретения
Задачей изобретения является разработка способа выявления событий редактирования гена ISA1 у зерновых культур, который был бы более простым в осуществлении и менее трудоемким и финансово затратным.
Для решения этой задачи авторы предлагают использовать способ, основанный на проведении полимеразной цепной реакции (ПЦР) с олигонуклеотидными праймерами, фланкирующими сайт предполагаемого редактирования, с последующей детекцией результатов ПЦР с помощью капиллярного электрофореза.
Заявленный способ предусматривает использование набора олигонуклеотидных праймеров (олигонуклеотидов), которые фланкируют три сайта редактирования гена ISA1 (Фигура 1). Нуклеотидные последовательности этих праймеров приведены ниже в Таблице 1 и в Перечне последовательностей.

Способ осуществляется путем проведения полимеразной цепной реакции с представленными в Таблице 1 олигонуклеотидами, визуализацией продукта с помощью капиллярного электрофореза и дальнейшем сравнении размера фрагментов контрольного образца (растение, которое не подвергалось редактированию) и редактированных растений. Благодаря точному определению длины амплифицированных фрагментов на капиллярном электрофорезе, сдвиг -разница в размере контрольного и исследуемых образцов показывает наличие редактирования у исследуемых растений и размер инсерций и делеций.
Краткое описание чертежей
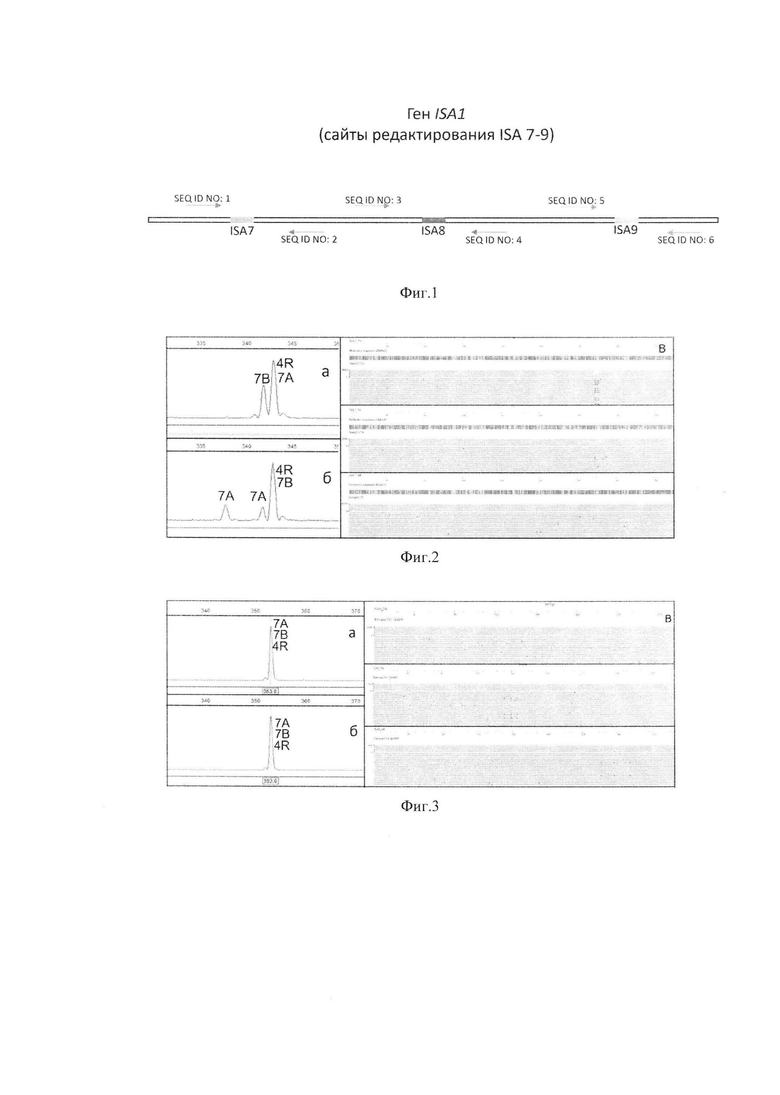
Фигура 1. Расположение сайтов редактирования и олигонуклеотидов, используемых для выявления редактирования в этих сайтах на гене ISA1
Фигура 2. Фореграммы образцов тритикале с парой олигонуклеотидов SEQ ID NO: 1 - SEQ ID NO: 2 для выявления генетических модификаций в сайте редактирования ISA7 гена ISA1
а) фореграмма контрольного (немодифицированного) образца
б) фореграмма исследуемого образца - выявлено редактирование: хромосома 7А - делеция 4 bp (40%), делеция 5 bp (40%) и редактирования нет (20%), 7В - редактирования нет, 4R - редактирования нет
в) картирование ридов исследуемого образца на референсную немодифицированную последовательность образца - выявлено редактирование: хромосома 7А - делеция 4 bp (40%), делеция 5 bp (40%) и редактирования нет (20%), 7В - редактирования нет, 4R - редактирования нет.
Фигура 3. Фореграммы образцов тритикале с парой олигонуклеотидов SEQ ID NO: 3 - SEQ ID NO: 4 для выявления генетических модификаций в сайте редактирования ISA8 гена ISA1
а) фореграмма контрольного (немодифицированного) образца
б) фореграмма исследуемого образца - редактирование не выявлено
в) картирование ридов исследуемого образца на референсную немодифицированную последовательность - редактирование не выявлено
Фигура 4. Фореграммы образцов тритикале с парой олигонуклеотидов SEQ ID NO: 5 - SEQ ID NO: 6 для выявления генетических модификаций в сайте редактирования ISA9 гена ISA1
а) форезграмма контрольного (немодифицированного) образца
б) фореграмма исследуемого образца - выявлено редактирование: хромосома 7А - делеция 4 bp, 7В - редактирования нет, 4R - редактирования нет
в) картирование ридов исследуемого образца на референсную немодифицированную последовательность - редактирование не выявлено
Фигура 5. Фореграммы образцов твердой пшеницы с парой олигонуклеотидов SEQ ID NO: 1 - SEQ ID NO: 2 для выявления генетических модификаций в сайте редактирования ISA7 гена ISA1
а) фореграмма контрольного (немодифицированного) образца
б) фореграмма исследуемого образца - выявлено редактирование: хромосома 7А - делеция 4 bp (40%) и редактирования нет (60%), 7В - делеция I bp (40%), инсерция 1 bp (40%) и редактирования нет (20%).
в) картирование ридов исследуемого образца на референсную немодифицированную последовательность - выявлено редактирование: хромосома 7А - делеция 4 bp (40%) и редактирования нет (60%), 7В - делеция 1 bp (40%), инсерция 1 bp (40%) и редактирования нет (20%).
Фигура 6. Фореграммы образцов твердой пшеницы с парой олигонуклеотидов SEQ ID NO: 3 - SEQ ID NO: 4 для выявления генетических модификаций в сайте редактирования ISA8 гена ISA 1
а) фореграмма контрольного (немодифицированного) образца
б) фореграмма исследуемого образца - выявлено редактирование: хромосома 7А - редактирования нет, 7В - инсерция 1 bp (35%) и редактирования нет (65%)
в) картирование ридов исследуемого образца на референсную немодифицированную последовательность - выявлено редактирование: хромосома 7А - редактирования нет, 7В - инсерция 1 bp (35%) и редактирования нет (65%).
Фигура 7. Фореграммы образцов твердой пшеницы с парой олигонуклеотидов SEQ ID NO: 5 - SEQ ID NO: 6 для выявления генетических модификаций в сайте редактирования ISA9 гена ISA1
а) форезграмма контрольного (немодифицированного) образца
б) фореграмма исследуемого образца - редактирование не выявлено
в) картирование ридов исследуемого образца на референсную немодифицированную последовательность - редактирование не выявлено
Осуществление изобретения (Примеры)
Настоящее изобретение было валидировано на растениях являющихся аллополиплоидами, а именно яровой и озимой тритикале и твердой пшеницы, имеющих генотип с различающимися субгеномами (ABR в случае тритикале и АВ в случае твердой пшеницы).
Пример 1
На первом этапе проводилось выделение ДНК из 78 растений-регенерантов озимой и яровой тритикале прошедших редактирование гена ISA1. Выделение проводили из сухих листьев СТАВ-методом (Springer et al, 2010) модифицированным для зерновых культур. Для дальнейшей постановки ПЦР олигонуклеотиды использовались в следующих комбинациях: SEQ ID NO: 1-SEQ ID NO: 2; SEQ ID NO: 3 - SEQ ID NO: 4; SEQ ID NO: 5 - SEQ ID NO: 6.
ПЦР-амплификацию проводили по единому режиму для всех пар олигонуклеотидов (95°С - 3 мин; 95°С - 20 сек, 60°С - 30 сек, 72°С - 20 сек - 35 циклов; 72°С - 10 мин), в стандартной ПЦР-смеси (Mg2+ 2.5 мМ, dNTP 0.25 мМ) с добавлением 1 мкл ДНК с концентрацией 5-10 нг/мкл. При постановке реакций в реакционную смесь так же добавляли 0.5 мкл ДМСО 100% для лучшей амплификации GC-богатых регионов. Продукты амплификации с ДМСО перед капиллярным форезом разводили дистиллированной водой в 20 раз с целью снижения концентрации солей для лучшего прохождения электрофореза.
Продукты амплификации визуализировали методом капиллярного электрофореза с помощью генетического анализатора Нанофор 05 в присутствии маркера молекулярного веса. Капиллярный электрофорез на генетическом анализаторе проводится в соответствии с руководством пользователя, предоставляемым его производителем (Синтол, Россия). Параметры электрофореза зависели от длины капилляров и типа полимера, рекомендуемые параметры инжекции 1800 В, 5 секунд. Продукты амлификации, полученные в постановке ПЦР с олигонуклеотидами 1) SEQ ID NO: 1-2; 2) SEQ ID NO: 3-4; 3) SEQ ID NO: 5-6 визуализировали на фрагментном анализе одновременно.
Результаты постановок - получившиеся профили образцов, проходившие редактирование, сравнивали с профилем контрольного образца, не проходившего редактирование и за счет изменения размера фрагментов выявляли редактирование и его тип.
Далее все образцы были отправлены на NGS. Библиотеки для NGS были подготовлены по протоколу 16S Metagenomic Sequencing Library Preparation (Preparing 16S Ribosomal RNA Gene Amplicons for the Illumina MiSeq System). Секвенирование проводилось на приборе Illumina MiSeq.
Для каждого секвенируемого образца длина прочтения, качество и количество ридов в полученных fastq-файлах оценивались с помощью программы FastQC 0.11.9. Первичная обработка ридов осуществлялась с помощью программы Trimmomatic-0.39. После тримминга fastq файлы конвертировались в fasta файлы, которые анализировались в программе Unipro UGENE 33.0.
Полученные результаты фрагментного анализа совпали с результатами NGS в 100% случаев (Фигуры 2-4), что демонстрирует высокую эффективность и надежность метода в выявлении генетических изменений, данные результаты также подчеркивают перспективность и потенциал данного метода и его дальнейшего использования.
Пример 2
Данный набор олигонуклеотидов также был апробирован на 68 растениях твердой пшеницы, где продемонстрировал высокую эффективность и точность при выявлении редактирования. Выделение ДНК, ПЦР-амплификацию, постановку фрагментного анализа и NGS проводили аналогично Примеру 1. Полученные результаты фрагментного анализа совпали с результатами NGS в 100% случаев, чем подтверждают возможность его использования для быстрого обнаружения генетических модификаций в твердой пшенице и, вероятно, в других ценных сельскохозяйственных культурах (Фигуры 5-7).
Финансирование работ по созданию настоящего изобретения проводилось из средств Соглашения №075-15-2019-1667 от «31» октября 2019 г. о предоставлении из федерального бюджета грантов в форме субсидий в соответствии с пунктом 4 статьи 78.1 Бюджетного кодекса Российской Федерации на осуществление государственной поддержки создания и развития центра геномных исследований мирового уровня «Курчатовский геномный центр» в рамках реализации федерального проекта «Развитие научной и научно-производственной кооперации» национального проекта «Наука».
Список литературы
Burton et al. (1 July 2002 г.). Starch granule initiation and growth are altered in barley mutants that lack isoamylase activity. The plant journal, p. 97-112.
Carmo C.D. (15 April 2020 г.). Identification and validation of mutation points associated with waxy phenotype in cassava. BMC Plant Biology, p. 164.
Ferreira et al. (14 July 2017 г.). Simultaneous silencing of isoamylases ISA1, ISA2 and ISA3 by multi-target RNAi in potato tubers leads to decreased starch content and an early sprouting phenotype. Plos one.
James et al. (1 April 1995 г.). Characterization of the maize gene sugary 1, a determinant of starch composition in kernels. The Plant Cell, p. 417-429.
Kubo et al. (1 January 2005 г.). Complementation of sugary-1 Phenotype in Rice Endosperm with the Wheat Isoamylase 1 Gene Supports a Direct Role for Isoamylase 1 in Amylopectin Biosynthesis. Plant Physiology, p. 43-56.
Kubo et al. (2 October 1999 г.). The Starch-Debranching Enzymes Isoamylase and Pullulanase Are Both Involved in Amylopectin Biosynthesis in Rice Endosperm. Plant Physiology, p. 399-410.
Pérez L. at al. (4 February 2019 г. ). CRISPR/Cas9 mutations in the rice Waxy/ISA1 gene induce allele-specific and zygosity-dependent feedback effects on endosperm starch biosynthesis. Plant Cell Reports, p. 417-433.
Pfister B. and Zeeman S.C. (11 May 2016 г.). Formation of starch in plant cells. Cellular and Molecular Life Sciences.
Sestili et al. (November 2016 г.). The down-regulation of the genes encoding Isoamylase 1 alters the starch composition of the durum wheat grain. Plant Science, p. 230-238.
Springer et al. (November 2010 г.). Isolation of Plant DNA for PCR and Genotyping Using Organic Extraction and СТАВ. Cold Spring Harbor Protocols.
Thompson, D. (November 2000 г.). On the non-random nature of amylopectin branching. Carbohydrate Polymers, p. 223-239.
Wattebled et al. (1 May 2005 г.). Mutants of Arabidopsis Lacking a Chloroplastic Isoamylase Accumulate Phytoglycogen and an Abnormal Form of Amylopectin. Plant Physiology, p. 184-195.
Wenzhi J. at al. (1 November 2013 г.). Demonstration of CRISPR/Cas9/sgRNA-mediated targeted gene modification in Arabidopsis, tobacco, sorghum and rice. Nucleic Acids Research, p. 188.
Zhang S. at al. (26 November 2018 г.). Targeted mutagenesis using the Agrobacterium tumefaciens-mediated CRISPR-Cas9 system in common wheat. BMC Plant Biology, p. 302.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="Перечень
последовательностей ISA1-1.xml" softwareName="WIPO Sequence"
softwareVersion="2.1.2" productionDate="2023-11-23">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>ISA1-1</ApplicationNumberText>
<FilingDate></FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>ISA1-1</ApplicantFileReference>
<ApplicantName languageCode="ru">ФГБНУ ВНИИСБ</ApplicantName>
<ApplicantNameLatin>All-Russia Research Institute of Agricultural
Biotechnology</ApplicantNameLatin>
<InventionTitle languageCode="ru">СПОСОБ ВЫЯВЛЕНИЯ СОБЫТИЙ
РЕДАКТИРОВАНИЯ ГЕНА ISA1 У ЗЕРНОВЫХ КУЛЬТУР С ПОМОЩЬЮ НАБОРА
ОЛИГОНУКЛЕОТИДНЫХ ПОСЛЕДОВАТЕЛЬНОСТЕЙ</InventionTitle>
<SequenceTotalQuantity>6</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttcacgccagaagatctcaag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>accaagatgcaggtaaagctaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q3">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>taytgtcttgccatctccagac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcttacctcaattccccgtttg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q5">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cataatgaccagaggttccaggt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcctccaagaattgggtcattg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| СПОСОБ ВЫЯВЛЕНИЯ СОБЫТИЙ РЕДАКТИРОВАНИЯ ГЕНА ISA1 У ЗЕРНОВЫХ КУЛЬТУР С ПОМОЩЬЮ НАБОРА ОЛИГОНУКЛЕОТИДНЫХ ПОСЛЕДОВАТЕЛЬНОСТЕЙ | 2023 |
|
RU2833966C1 |
| СПОСОБ ВЫЯВЛЕНИЯ СОБЫТИЙ РЕДАКТИРОВАНИЯ ГЕНА GBSSI У ЗЕРНОВЫХ КУЛЬТУР С ПОМОЩЬЮ НАБОРА ОЛИГОНУКЛЕОТИДНЫХ ПОСЛЕДОВАТЕЛЬНОСТЕЙ | 2023 |
|
RU2834229C1 |
| СПОСОБ ВЫЯВЛЕНИЯ СОБЫТИЙ РЕДАКТИРОВАНИЯ ГЕНА SBEIIA У ЗЕРНОВЫХ КУЛЬТУР С ПОМОЩЬЮ НАБОРА ОЛИГОНУКЛЕОТИДНЫХ ПОСЛЕДОВАТЕЛЬНОСТЕЙ | 2023 |
|
RU2833965C1 |
| СПОСОБ ВЫЯВЛЕНИЯ СОБЫТИЙ РЕДАКТИРОВАНИЯ ГЕНА RSR1 У ЗЕРНОВЫХ КУЛЬТУР С ПОМОЩЬЮ НАБОРА ОЛИГОНУКЛЕОТИДНЫХ ПОСЛЕДОВАТЕЛЬНОСТЕЙ | 2023 |
|
RU2833967C1 |
| СПОСОБ ВЫЯВЛЕНИЯ СОБЫТИЙ РЕДАКТИРОВАНИЯ ГЕНА RSR1 У ЗЕРНОВЫХ КУЛЬТУР С ПОМОЩЬЮ НАБОРА ОЛИГОНУКЛЕОТИДНЫХ ПОСЛЕДОВАТЕЛЬНОСТЕЙ | 2023 |
|
RU2833964C1 |
| СПОСОБ ВЫЯВЛЕНИЯ СОБЫТИЙ РЕДАКТИРОВАНИЯ ГЕНА GBSSI У ЗЕРНОВЫХ КУЛЬТУР С ПОМОЩЬЮ НАБОРА ОЛИГОНУКЛЕОТИДНЫХ ПОСЛЕДОВАТЕЛЬНОСТЕЙ | 2023 |
|
RU2817377C1 |
| СПОСОБ ВЫЯВЛЕНИЯ СОБЫТИЙ РЕДАКТИРОВАНИЯ ГЕНА SSIIA У ЗЕРНОВЫХ КУЛЬТУР С ПОМОЩЬЮ НАБОРА ОЛИГОНУКЛЕОТИДНЫХ ПОСЛЕДОВАТЕЛЬНОСТЕЙ | 2023 |
|
RU2839687C1 |
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ КАРТОФЕЛЯ С ТРИАЛЛЕЛЬНЫМИ МУТАЦИЯМИ В КОДИРУЮЩЕЙ ОБЛАСТИ ГЕНА EDR1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2022 |
|
RU2817378C2 |
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ КАРТОФЕЛЯ С ТЕТРААЛЛЕЛЬНЫМИ МУТАЦИЯМИ В КОДИРУЮЩЕЙ ОБЛАСТИ ГЕНА EDR1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2022 |
|
RU2817384C2 |
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ КАРТОФЕЛЯ С ТЕТРААЛЛЕЛЬНЫМИ МУТАЦИЯМИ В КОДИРУЮЩЕЙ ОБЛАСТИ ГЕНА EDR1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2022 |
|
RU2800828C1 |


Настоящее изобретение относится к области биотехнологии и генной инженерии растений, а именно к генетическим маркерам, выявляющим генетические модификации гена ISA1, полученные с использованием CRISPR/Cas9 на геноме твердой пшеницы (Triticum durum) и озимой и яровой тритикале (×Triticosecale). Раскрыт способ, основанный на проведении полимеразной цепной реакции (ПЦР) с олигонуклеотидными праймерами, фланкирующими сайт предполагаемого редактирования, с последующей детекцией результатов ПЦР с помощью капиллярного электрофореза. Изобретение позволяет выявлять события редактирования гена ISA1 у зерновых культур, возникающие в результате редактирования с помощью системы CRISPR/Cas9. 2 з.п. ф-лы, 7 ил., 1 табл., 2 пр.
1. Способ выявления событий редактирования в сайтах редактирования ISA7, ISA8 и ISA9 гена ISA1 у зерновых культур, включающий в себя следующие этапы:
выделение геномной ДНК, параллельная постановка ПЦР-амплификации со следующими парами олигонуклеотидных последовательностей SEQ ID NO: 1 - SEQ ID NO: 2; SEQ ID NO: 3 - SEQ ID NO: 4; SEQ ID NO: 5 - SEQ ID NO: 6, визуализация при помощи капиллярного электрофореза, сравнение подвижности продуктов амплификации исследуемого и контрольного образца, полученного от растения, которое не подвергалось редактированию, и установление события редактирования в случае различия подвижности исследуемого и контрольного образца.
2. Способ по п. 1, отличающийся тем, что зерновой культурой является пшеница.
3. Способ по п. 1, отличающийся тем, что зерновой культурой является тритикале.
| Участки генов и гены, ассоциированные с повышенной урожайностью у растений | 2016 |
|
RU2758718C2 |
| СПОСОБ ПРОВЕДЕНИЯ ПЦР И ПЦР-ПДРФ ДЛЯ ИДЕНТИФИКАЦИИ АЛЛЕЛЬНЫХ ВАРИАНТОВ Waxy-ГЕНОВ ПШЕНИЦЫ | 2013 |
|
RU2528748C1 |
| US 10557146 B2, 11.02.2020 | |||
| Крупин П.Ю | |||
| Выбор генов синтеза крахмала пшеницы как мишеней для геномного редактирования / П | |||
| Ю | |||
| Крупин // Биотехнология в растениеводстве, животноводстве и сельскохозяйственной микробиологии: Сборник тезисов докладов 20-й Всероссийской конференции молодых учёных, | |||
Авторы
Даты
2025-02-03—Публикация
2023-12-01—Подача